KR20150016588A - Rna-유도된 표적 dna 변형 및 전사의 rna-유도된 조절을 위한 방법 및 조성물 - Google Patents
Rna-유도된 표적 dna 변형 및 전사의 rna-유도된 조절을 위한 방법 및 조성물 Download PDFInfo
- Publication number
- KR20150016588A KR20150016588A KR1020147036096A KR20147036096A KR20150016588A KR 20150016588 A KR20150016588 A KR 20150016588A KR 1020147036096 A KR1020147036096 A KR 1020147036096A KR 20147036096 A KR20147036096 A KR 20147036096A KR 20150016588 A KR20150016588 A KR 20150016588A
- Authority
- KR
- South Korea
- Prior art keywords
- dna
- activity
- rna
- site
- cells
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Ceased
Links
- 238000000034 method Methods 0.000 title claims abstract 95
- 238000013518 transcription Methods 0.000 title claims abstract 32
- 230000035897 transcription Effects 0.000 title claims abstract 32
- 239000000203 mixture Substances 0.000 title claims abstract 21
- 230000008836 DNA modification Effects 0.000 title 1
- 108020004414 DNA Proteins 0.000 claims abstract 122
- 108090000765 processed proteins & peptides Proteins 0.000 claims abstract 111
- 229920001184 polypeptide Polymers 0.000 claims abstract 108
- 102000004196 processed proteins & peptides Human genes 0.000 claims abstract 108
- 108091032973 (ribonucleotides)n+m Proteins 0.000 claims abstract 95
- 108091033409 CRISPR Proteins 0.000 claims abstract 24
- 102000039446 nucleic acids Human genes 0.000 claims abstract 19
- 108020004707 nucleic acids Proteins 0.000 claims abstract 19
- 150000007523 nucleic acids Chemical class 0.000 claims abstract 19
- 230000004048 modification Effects 0.000 claims abstract 3
- 238000012986 modification Methods 0.000 claims abstract 3
- 230000000694 effects Effects 0.000 claims 143
- 210000004027 cell Anatomy 0.000 claims 98
- 239000002773 nucleotide Substances 0.000 claims 80
- 125000003729 nucleotide group Chemical group 0.000 claims 80
- 125000003275 alpha amino acid group Chemical group 0.000 claims 43
- 108091033319 polynucleotide Proteins 0.000 claims 43
- 102000040430 polynucleotide Human genes 0.000 claims 43
- 239000002157 polynucleotide Substances 0.000 claims 43
- 102000004190 Enzymes Human genes 0.000 claims 36
- 108090000790 Enzymes Proteins 0.000 claims 36
- 230000000295 complement effect Effects 0.000 claims 30
- 239000013604 expression vector Substances 0.000 claims 22
- 230000004570 RNA-binding Effects 0.000 claims 20
- 238000003259 recombinant expression Methods 0.000 claims 20
- 230000035772 mutation Effects 0.000 claims 16
- 230000001747 exhibiting effect Effects 0.000 claims 13
- 108060004795 Methyltransferase Proteins 0.000 claims 12
- 101710163270 Nuclease Proteins 0.000 claims 12
- 239000003153 chemical reaction reagent Substances 0.000 claims 12
- 101000615488 Homo sapiens Methyl-CpG-binding domain protein 2 Proteins 0.000 claims 10
- 102100021299 Methyl-CpG-binding domain protein 2 Human genes 0.000 claims 10
- 108091028043 Nucleic acid sequence Proteins 0.000 claims 10
- 102000016397 Methyltransferase Human genes 0.000 claims 9
- 210000004102 animal cell Anatomy 0.000 claims 9
- 230000002255 enzymatic effect Effects 0.000 claims 9
- 108010033040 Histones Proteins 0.000 claims 8
- 150000001413 amino acids Chemical class 0.000 claims 8
- 101150106478 GPS1 gene Proteins 0.000 claims 7
- 230000005782 double-strand break Effects 0.000 claims 7
- 102000004160 Phosphoric Monoester Hydrolases Human genes 0.000 claims 6
- 108090000608 Phosphoric Monoester Hydrolases Proteins 0.000 claims 6
- 108091000080 Phosphotransferase Proteins 0.000 claims 6
- 241000283984 Rodentia Species 0.000 claims 6
- 102000005421 acetyltransferase Human genes 0.000 claims 6
- 108020002494 acetyltransferase Proteins 0.000 claims 6
- 239000000872 buffer Substances 0.000 claims 6
- 238000010790 dilution Methods 0.000 claims 6
- 239000012895 dilution Substances 0.000 claims 6
- 210000003527 eukaryotic cell Anatomy 0.000 claims 6
- 230000035876 healing Effects 0.000 claims 6
- 210000004962 mammalian cell Anatomy 0.000 claims 6
- 102000020233 phosphotransferase Human genes 0.000 claims 6
- 230000001105 regulatory effect Effects 0.000 claims 6
- 241000251468 Actinopterygii Species 0.000 claims 5
- 241000251539 Vertebrata <Metazoa> Species 0.000 claims 5
- 230000001580 bacterial effect Effects 0.000 claims 5
- 210000004602 germ cell Anatomy 0.000 claims 5
- 210000001082 somatic cell Anatomy 0.000 claims 5
- 210000000130 stem cell Anatomy 0.000 claims 5
- 102220605874 Cytosolic arginine sensor for mTORC1 subunit 2_D10A_mutation Human genes 0.000 claims 4
- 108010053770 Deoxyribonucleases Proteins 0.000 claims 4
- 102000016911 Deoxyribonucleases Human genes 0.000 claims 4
- 241000700159 Rattus Species 0.000 claims 4
- 241000193996 Streptococcus pyogenes Species 0.000 claims 4
- 230000027455 binding Effects 0.000 claims 4
- 102000004169 proteins and genes Human genes 0.000 claims 4
- 241000283707 Capra Species 0.000 claims 3
- 230000005778 DNA damage Effects 0.000 claims 3
- 231100000277 DNA damage Toxicity 0.000 claims 3
- 102100034343 Integrase Human genes 0.000 claims 3
- 108010061833 Integrases Proteins 0.000 claims 3
- 102000003960 Ligases Human genes 0.000 claims 3
- 108090000364 Ligases Proteins 0.000 claims 3
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 claims 3
- 239000004472 Lysine Substances 0.000 claims 3
- 241000699666 Mus <mouse, genus> Species 0.000 claims 3
- 241001494479 Pecora Species 0.000 claims 3
- CZPWVGJYEJSRLH-UHFFFAOYSA-N Pyrimidine Chemical compound C1=CN=CN=C1 CZPWVGJYEJSRLH-UHFFFAOYSA-N 0.000 claims 3
- 108091093078 Pyrimidine dimer Proteins 0.000 claims 3
- 102000008579 Transposases Human genes 0.000 claims 3
- 108010020764 Transposases Proteins 0.000 claims 3
- 102000006275 Ubiquitin-Protein Ligases Human genes 0.000 claims 3
- 108010083111 Ubiquitin-Protein Ligases Proteins 0.000 claims 3
- 230000001154 acute effect Effects 0.000 claims 3
- 230000015572 biosynthetic process Effects 0.000 claims 3
- 210000000349 chromosome Anatomy 0.000 claims 3
- 238000011033 desalting Methods 0.000 claims 3
- 210000005260 human cell Anatomy 0.000 claims 3
- 238000000338 in vitro Methods 0.000 claims 3
- 238000001727 in vivo Methods 0.000 claims 3
- 230000001939 inductive effect Effects 0.000 claims 3
- 230000007498 myristoylation Effects 0.000 claims 3
- 230000001590 oxidative effect Effects 0.000 claims 3
- 108090000623 proteins and genes Proteins 0.000 claims 3
- 239000013635 pyrimidine dimer Substances 0.000 claims 3
- OSXFATOLZGZLSK-UHFFFAOYSA-N 6,7-dimethoxy-2-(4-methyl-1,4-diazepan-1-yl)-N-[1-(phenylmethyl)-4-piperidinyl]-4-quinazolinamine Chemical compound C=12C=C(OC)C(OC)=CC2=NC(N2CCN(C)CCC2)=NC=1NC(CC1)CCN1CC1=CC=CC=C1 OSXFATOLZGZLSK-UHFFFAOYSA-N 0.000 claims 2
- 108010016918 Histone-Lysine N-Methyltransferase Proteins 0.000 claims 2
- 102000000581 Histone-lysine N-methyltransferase Human genes 0.000 claims 2
- 102000007077 Lysine Acetyltransferases Human genes 0.000 claims 2
- 108010033293 Lysine Acetyltransferases Proteins 0.000 claims 2
- 108091005461 Nucleic proteins Proteins 0.000 claims 2
- 102000055027 Protein Methyltransferases Human genes 0.000 claims 2
- 108700040121 Protein Methyltransferases Proteins 0.000 claims 2
- 239000012190 activator Substances 0.000 claims 2
- 230000009615 deamination Effects 0.000 claims 2
- 238000006481 deamination reaction Methods 0.000 claims 2
- 239000000839 emulsion Substances 0.000 claims 2
- 230000009466 transformation Effects 0.000 claims 2
- 230000009261 transgenic effect Effects 0.000 claims 2
- 239000011534 wash buffer Substances 0.000 claims 2
- 102000040650 (ribonucleotides)n+m Human genes 0.000 claims 1
- SWWQQSDRUYSMAR-UHFFFAOYSA-N 1-[(4-hydroxyphenyl)methyl]-1,2,3,4-tetrahydroisoquinoline-6,7-diol;hydrochloride Chemical group Cl.C1=CC(O)=CC=C1CC1C2=CC(O)=C(O)C=C2CCN1 SWWQQSDRUYSMAR-UHFFFAOYSA-N 0.000 claims 1
- 241000269350 Anura Species 0.000 claims 1
- 241000203069 Archaea Species 0.000 claims 1
- 241000271566 Aves Species 0.000 claims 1
- 241000894006 Bacteria Species 0.000 claims 1
- 230000007067 DNA methylation Effects 0.000 claims 1
- 102100035102 E3 ubiquitin-protein ligase MYCBP2 Human genes 0.000 claims 1
- 241000206602 Eukaryota Species 0.000 claims 1
- 241000124008 Mammalia Species 0.000 claims 1
- 241001465754 Metazoa Species 0.000 claims 1
- 241000699670 Mus sp. Species 0.000 claims 1
- 108700019146 Transgenes Proteins 0.000 claims 1
- 238000003776 cleavage reaction Methods 0.000 claims 1
- 238000010367 cloning Methods 0.000 claims 1
- 238000010168 coupling process Methods 0.000 claims 1
- 238000005859 coupling reaction Methods 0.000 claims 1
- 230000006240 deamidation Effects 0.000 claims 1
- 238000006114 decarboxylation reaction Methods 0.000 claims 1
- 230000029087 digestion Effects 0.000 claims 1
- 238000009499 grossing Methods 0.000 claims 1
- 239000003112 inhibitor Substances 0.000 claims 1
- 230000004807 localization Effects 0.000 claims 1
- 238000004519 manufacturing process Methods 0.000 claims 1
- 239000008267 milk Substances 0.000 claims 1
- 210000004080 milk Anatomy 0.000 claims 1
- 235000013336 milk Nutrition 0.000 claims 1
- 230000002093 peripheral effect Effects 0.000 claims 1
- 238000006303 photolysis reaction Methods 0.000 claims 1
- 230000015843 photosynthesis, light reaction Effects 0.000 claims 1
- 210000001236 prokaryotic cell Anatomy 0.000 claims 1
- 230000001737 promoting effect Effects 0.000 claims 1
- 230000037425 regulation of transcription Effects 0.000 claims 1
- 230000022532 regulation of transcription, DNA-dependent Effects 0.000 claims 1
- 230000007017 scission Effects 0.000 claims 1
- 230000010741 sumoylation Effects 0.000 claims 1
- 230000008685 targeting Effects 0.000 claims 1
- 230000034512 ubiquitination Effects 0.000 claims 1
- 238000010798 ubiquitination Methods 0.000 claims 1
Images
Classifications
-
- A—HUMAN NECESSITIES
- A01—AGRICULTURE; FORESTRY; ANIMAL HUSBANDRY; HUNTING; TRAPPING; FISHING
- A01H—NEW PLANTS OR NON-TRANSGENIC PROCESSES FOR OBTAINING THEM; PLANT REPRODUCTION BY TISSUE CULTURE TECHNIQUES
- A01H6/00—Angiosperms, i.e. flowering plants, characterised by their botanic taxonomy
- A01H6/46—Gramineae or Poaceae, e.g. ryegrass, rice, wheat or maize
- A01H6/4684—Zea mays [maize]
-
- A—HUMAN NECESSITIES
- A01—AGRICULTURE; FORESTRY; ANIMAL HUSBANDRY; HUNTING; TRAPPING; FISHING
- A01K—ANIMAL HUSBANDRY; AVICULTURE; APICULTURE; PISCICULTURE; FISHING; REARING OR BREEDING ANIMALS, NOT OTHERWISE PROVIDED FOR; NEW BREEDS OF ANIMALS
- A01K67/00—Rearing or breeding animals, not otherwise provided for; New or modified breeds of animals
- A01K67/027—New or modified breeds of vertebrates
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K38/00—Medicinal preparations containing peptides
- A61K38/16—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- A61K38/43—Enzymes; Proenzymes; Derivatives thereof
- A61K38/46—Hydrolases (3)
- A61K38/465—Hydrolases (3) acting on ester bonds (3.1), e.g. lipases, ribonucleases
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P31/00—Antiinfectives, i.e. antibiotics, antiseptics, chemotherapeutics
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P31/00—Antiinfectives, i.e. antibiotics, antiseptics, chemotherapeutics
- A61P31/04—Antibacterial agents
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P31/00—Antiinfectives, i.e. antibiotics, antiseptics, chemotherapeutics
- A61P31/12—Antivirals
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P43/00—Drugs for specific purposes, not provided for in groups A61P1/00-A61P41/00
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/10—Processes for the isolation, preparation or purification of DNA or RNA
- C12N15/102—Mutagenizing nucleic acids
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/111—General methods applicable to biologically active non-coding nucleic acids
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/113—Non-coding nucleic acids modulating the expression of genes, e.g. antisense oligonucleotides; Antisense DNA or RNA; Triplex- forming oligonucleotides; Catalytic nucleic acids, e.g. ribozymes; Nucleic acids used in co-suppression or gene silencing
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/70—Vectors or expression systems specially adapted for E. coli
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/74—Vectors or expression systems specially adapted for prokaryotic hosts other than E. coli, e.g. Lactobacillus, Micromonospora
- C12N15/746—Vectors or expression systems specially adapted for prokaryotic hosts other than E. coli, e.g. Lactobacillus, Micromonospora for lactic acid bacteria (Streptococcus; Lactococcus; Lactobacillus; Pediococcus; Enterococcus; Leuconostoc; Propionibacterium; Bifidobacterium; Sporolactobacillus)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/87—Introduction of foreign genetic material using processes not otherwise provided for, e.g. co-transformation
- C12N15/90—Stable introduction of foreign DNA into chromosome
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/87—Introduction of foreign genetic material using processes not otherwise provided for, e.g. co-transformation
- C12N15/90—Stable introduction of foreign DNA into chromosome
- C12N15/902—Stable introduction of foreign DNA into chromosome using homologous recombination
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/87—Introduction of foreign genetic material using processes not otherwise provided for, e.g. co-transformation
- C12N15/90—Stable introduction of foreign DNA into chromosome
- C12N15/902—Stable introduction of foreign DNA into chromosome using homologous recombination
- C12N15/907—Stable introduction of foreign DNA into chromosome using homologous recombination in mammalian cells
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N5/00—Undifferentiated human, animal or plant cells, e.g. cell lines; Tissues; Cultivation or maintenance thereof; Culture media therefor
- C12N5/10—Cells modified by introduction of foreign genetic material
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/14—Hydrolases (3)
- C12N9/16—Hydrolases (3) acting on ester bonds (3.1)
- C12N9/22—Ribonucleases [RNase]; Deoxyribonucleases [DNase]
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6844—Nucleic acid amplification reactions
- C12Q1/686—Polymerase chain reaction [PCR]
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y301/00—Hydrolases acting on ester bonds (3.1)
- C12Y301/04—Phosphoric diester hydrolases (3.1.4)
-
- H—ELECTRICITY
- H01—ELECTRIC ELEMENTS
- H01L—SEMICONDUCTOR DEVICES NOT COVERED BY CLASS H10
- H01L21/00—Processes or apparatus adapted for the manufacture or treatment of semiconductor or solid state devices or of parts thereof
- H01L21/02—Manufacture or treatment of semiconductor devices or of parts thereof
- H01L21/02104—Forming layers
- H01L21/02107—Forming insulating materials on a substrate
- H01L21/02296—Forming insulating materials on a substrate characterised by the treatment performed before or after the formation of the layer
- H01L21/02299—Forming insulating materials on a substrate characterised by the treatment performed before or after the formation of the layer pre-treatment
- H01L21/02312—Forming insulating materials on a substrate characterised by the treatment performed before or after the formation of the layer pre-treatment treatment by exposure to a gas or vapour
-
- H—ELECTRICITY
- H01—ELECTRIC ELEMENTS
- H01L—SEMICONDUCTOR DEVICES NOT COVERED BY CLASS H10
- H01L21/00—Processes or apparatus adapted for the manufacture or treatment of semiconductor or solid state devices or of parts thereof
- H01L21/02—Manufacture or treatment of semiconductor devices or of parts thereof
- H01L21/02104—Forming layers
- H01L21/02365—Forming inorganic semiconducting materials on a substrate
-
- H—ELECTRICITY
- H10—SEMICONDUCTOR DEVICES; ELECTRIC SOLID-STATE DEVICES NOT OTHERWISE PROVIDED FOR
- H10H—INORGANIC LIGHT-EMITTING SEMICONDUCTOR DEVICES HAVING POTENTIAL BARRIERS
- H10H20/00—Individual inorganic light-emitting semiconductor devices having potential barriers, e.g. light-emitting diodes [LED]
- H10H20/01—Manufacture or treatment
- H10H20/011—Manufacture or treatment of bodies, e.g. forming semiconductor layers
- H10H20/013—Manufacture or treatment of bodies, e.g. forming semiconductor layers having light-emitting regions comprising only Group III-V materials
- H10H20/0137—Manufacture or treatment of bodies, e.g. forming semiconductor layers having light-emitting regions comprising only Group III-V materials the light-emitting regions comprising nitride materials
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K48/00—Medicinal preparations containing genetic material which is inserted into cells of the living body to treat genetic diseases; Gene therapy
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/70—Fusion polypeptide containing domain for protein-protein interaction
- C07K2319/71—Fusion polypeptide containing domain for protein-protein interaction containing domain for transcriptional activaation, e.g. VP16
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/85—Fusion polypeptide containing an RNA binding domain
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/10—Type of nucleic acid
- C12N2310/11—Antisense
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/10—Type of nucleic acid
- C12N2310/13—Decoys
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/10—Type of nucleic acid
- C12N2310/14—Type of nucleic acid interfering nucleic acids [NA]
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/10—Type of nucleic acid
- C12N2310/20—Type of nucleic acid involving clustered regularly interspaced short palindromic repeats [CRISPR]
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/30—Chemical structure
- C12N2310/31—Chemical structure of the backbone
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/30—Chemical structure
- C12N2310/32—Chemical structure of the sugar
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/30—Chemical structure
- C12N2310/33—Chemical structure of the base
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/30—Chemical structure
- C12N2310/35—Nature of the modification
- C12N2310/351—Conjugate
- C12N2310/3519—Fusion with another nucleic acid
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/50—Physical structure
- C12N2310/53—Physical structure partially self-complementary or closed
- C12N2310/531—Stem-loop; Hairpin
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2800/00—Nucleic acids vectors
- C12N2800/80—Vectors containing sites for inducing double-stranded breaks, e.g. meganuclease restriction sites
-
- Y—GENERAL TAGGING OF NEW TECHNOLOGICAL DEVELOPMENTS; GENERAL TAGGING OF CROSS-SECTIONAL TECHNOLOGIES SPANNING OVER SEVERAL SECTIONS OF THE IPC; TECHNICAL SUBJECTS COVERED BY FORMER USPC CROSS-REFERENCE ART COLLECTIONS [XRACs] AND DIGESTS
- Y02—TECHNOLOGIES OR APPLICATIONS FOR MITIGATION OR ADAPTATION AGAINST CLIMATE CHANGE
- Y02A—TECHNOLOGIES FOR ADAPTATION TO CLIMATE CHANGE
- Y02A50/00—TECHNOLOGIES FOR ADAPTATION TO CLIMATE CHANGE in human health protection, e.g. against extreme weather
- Y02A50/30—Against vector-borne diseases, e.g. mosquito-borne, fly-borne, tick-borne or waterborne diseases whose impact is exacerbated by climate change
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Genetics & Genomics (AREA)
- Engineering & Computer Science (AREA)
- Chemical & Material Sciences (AREA)
- Organic Chemistry (AREA)
- Biomedical Technology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Zoology (AREA)
- Wood Science & Technology (AREA)
- Biotechnology (AREA)
- General Engineering & Computer Science (AREA)
- Molecular Biology (AREA)
- General Health & Medical Sciences (AREA)
- Biochemistry (AREA)
- Microbiology (AREA)
- Physics & Mathematics (AREA)
- Biophysics (AREA)
- Plant Pathology (AREA)
- Medicinal Chemistry (AREA)
- Animal Behavior & Ethology (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Public Health (AREA)
- Veterinary Medicine (AREA)
- Pharmacology & Pharmacy (AREA)
- Mycology (AREA)
- Nuclear Medicine, Radiotherapy & Molecular Imaging (AREA)
- General Chemical & Material Sciences (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Environmental Sciences (AREA)
- Cell Biology (AREA)
- Communicable Diseases (AREA)
- Oncology (AREA)
- Immunology (AREA)
- Crystallography & Structural Chemistry (AREA)
- Epidemiology (AREA)
- Gastroenterology & Hepatology (AREA)
- Biodiversity & Conservation Biology (AREA)
- Animal Husbandry (AREA)
- Physiology (AREA)
Abstract
Description
도 2는 Cas9/Csn1 부위 지향적 변형 폴리펩타이드 및 DNA-표적화 RNA을 사용하여 도입된 이중-가닥 DNA 절단을 통한 표적 DNA 편집을 묘사한다.
도 3a-b는 스트렙토코쿠스 파이오제네스(서열번호 8)로부터 Cas9/Csn1 단백질의 아미노산 서열을 묘사한다. Cas9는 HNH 및 RuvC 엔도뉴클레아제 모두에 대해 상동인 도메인을 갖는다. (A) 모티프 1-4는 윗줄치고, (B) 도메인 1 및 2는 윗줄친다.
도 4a-b는 다중 종으로부터 Cas9/Csn1 단백질 사이의 퍼센트 동일성을 묘사한다. (A) 스트렙토코쿠스 파이오제네스에 대한 서열 동일성. 예를 들면, 도메인 1은 도 3B에 묘사된 스트렙토코쿠스 파이오제네스로부터의 Cas9/Csn1의 아미노산 7-166이고, 도메인 2는 아미노산 731-1003이다. (B) 나이세리아 메닌자이티디스(Neisseria meningitidis)에 대한 서열 동일성. 예를 들면, 도메인 1은 나이세리아 메닌자이티디스 (서열번호 79)로부터의 Cas9/Csn1의 아미노산 13-139이고, 도메인 2는 아미노산 475-750이다.
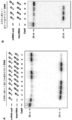
도 5는 도 32의 계통발생 표로부터 선택된 다양한 여러가지 종으로부터 Cas9/Csn1 단백질의 모티프 1-4의 다중 서열 정렬을 묘사한다(참고: 도 32, 도 3a 및 표 1)(스트렙토코쿠스 파이오제네스 (서열번호 8), 레지오넬라 뉴모필라( Legionella pneumophila )(서열번호 17), 감마 프로테오박테리움( Gamma proteobacterium)(서열번호 107), 리스테리아 이노쿠아( Listeria innocua )(서열번호 3), 락토바실러스 가쎄리(Lactobacillus gasseri)(서열번호 152), 유박테륨 렉탈레(Eubacterium rectale)(서열번호 99), 스타필로코쿠스 루그두넨시스(Staphylococcus lugdunensis)(서열번호 185), 마이코플라스마 사이노비애(Mycoplasma synoviae)(서열번호 22), 마이코플라스마 모빌레(Mycoplasma mobile)(서열번호 16), 울리넬라 석시노게네스(Wolinella succinogenes)(서열번호 10), 플라보박테리아 칼럼나레(Flavobacterium columnare)(서열번호 235), 파이브로박터 석시노게네스(Fibrobacter succinogenes)(서열번호 121), 박테로이데스 프라길리스(Bacteroides fragilis)(서열번호 21), 악시도테르무스 셀루로라이티쿠스(Acidothermus cellulolyticus)(서열번호 42) 및 비피도박테리움 덴티움(Bifidobacterium dentium)(서열번호 131).
도 6a-b는 다양한 종으로부터 자연발생 tracrRNA("활성제-RNA") 서열(엘. 이노쿠아(innocua)(서열번호 268); 에스. 파이오제네스(pyogenes)(서열번호 267); 에스. 뮤탄스(mutans)(서열번호 269); 에스. 서모필러스1(thermophilus1)(서열번호 270); 엠. 모빌레(mobile)(서열번호 274); 엔. 메닌자이티데스(meningitides)(서열번호 272); 피. 멀토시다(multocida)(서열번호 273); 에스. 서모필러스2(thermophilus2)(서열번호 271); 및 에스. 파이오제네스(pyogenes)(서열번호 267)의 정렬을 제공한다. (A) 유사한 구조의 CRISPR/Cas 유전자 좌 및 크게 유사한 Cas9/Csn1 서열과 연관된 선택된 tracrRNA 오쏘로그 (AlignX, 벡터NTI 패키지, Invitrogen)의 다중 서열 정렬. 블랙 박스는 공유된 뉴클레오타이드를 나타낸다 (B) 상이한 구조의 CRISPR/Cas 유전자 좌 및 비-밀접하게 관련된 Cas9/Csn1 서열과 연관된 선택된 tracrRNA 오쏘로그 (AlignX, 벡터NTI 패키지, Invitrogen)의 다중 서열 정렬. 엔. 메닌자이티디스(meningitidis) 및 피. 멀토시다(multocida) tracrRNA 오쏘로그의 서열 유사성을 주시한다. 블랙 박스는 공유된 뉴클레오타이드를 나타낸다. 보다 예시적인 활성제-RNA 서열의 경우, 서열번호 431-562를 참조한다.
도 7a-b는 다양한 종으로부터 crRNA ("타겟터-RNA") 서열의 자연 발생 듀플렉스-형성 세그먼트(엘. 이노쿠아 (서열번호 //); 에스. 파이오제네스 (서열번호 //); 에스. 뮤탄스 (서열번호 //); 에스. 서모필러스1 (서열번호 //); 씨. 제주니(jejuni)(서열번호 //); 에스. 파이오제네스 (서열번호 //); 에프. 노비시다(novicida)(서열번호 //); 엠. 모빌레 (서열번호 //); 엔. 메닌자이티데스 (서열번호 //); 피. 멀토시다 (서열번호 //); 및 S. 서모필러스2 (서열번호 //)의 정렬을 제공한다. (A) 유사한 구조의 유전자 좌 및 크게 유사한 Cas9/Csn1 서열과 연관된 타겟터-RNA 서열(AlignX, 벡터NTI 패키지, Invitrogen)의 예시적인 듀플렉스-형성 세그먼트의 다중 서열 정렬. (B) 상이한 구조의 유전자 좌 및 다양한 Cas9 서열과 연관된 타겟터-RNA 서열(AlignX, 벡터NTI 패키지, Invitrogen)의 예시적인 듀플렉스-형성 세그먼트의 다중 서열 정렬. 블랙 박스는 공유된 뉴클레오타이드를 나타낸다. 보다 예시적인 듀플렉스-형성 세그먼트 타겟터-RNA 서열의 경우, 서열번호 563-679를 참조한다.
도 8은 상응하는 tracrRNA 오쏘로그 ("활성제-RNA")의 듀플렉스-형성 세그먼트과 함께 crRNA("타겟터-RNA")의 자연 발생 듀플렉스-형성 세그먼트의 하이브리드화의 개략도를 제공한다. 상부 서열, 타겟터-RNA; 하부 서열, 상응하는 활성제-RNA의 듀플렉스-형성 세그먼트. CRISPR 유전자 좌는 유형 II (Nmeni/CASS4) CRISPR/Cas 시스템에 속한다. 명명법은 CRISPR 데이타베이스 (CRISPR DB)에 따른다. 에스. 파이오제네스 (서열번호 // 및 //); 에스. 뮤탄스 (서열번호 // 및 //); 에스. 서모필러스1 (서열번호 // 및 //); 에스. 서모필러스2 (서열번호 // 및 //); 엘. 이노쿠아 (서열번호 // 및 //); 티. 덴티콜라(denticola)(서열번호 // 및 //); 엔. 메닌자이티데스 (서열번호 // 및 //); 에스. 고르도니이(gordonii)(서열번호 // 및 //); B. 비피덤(bifidum)(서열번호 // 및 //); L. 살리바리우스(salivarius)(서열번호 // 및 //); F. 툴라렌시스(tularensis)(서열번호 // 및 //); 및 엘. 뉴모필라(pneumophila)(서열번호 // 및 //). 일부 종은 각각 두 개의 유형 II CRISPR 유전자 좌를 함유한다는 것을 주시한다. 보다 예시적인 활성제-RNA 서열을 위해, 서열번호 431-562를 참조한다. 보다 예시적인 듀플렉스-형성 세그먼트 타겟터-RNA 서열을 위해, 서열번호 563-679을 참조한다.
도 9는 두 종으로부터 tracrRNA (활성제-RNA) 및 crRNA (타겟터-RNA) 서열의 예를 묘사한다. 교환성의 정도가 존재한다; 예를 들면, S.파이오제네스 Cas9/Csn1 단백질은 L.이노쿠아로부터 유도된 tracrRNA 및 crRNA와 기능성이다. (|)은 표준적 왓슨-크릭(Watson-Crick) 염기쌍을 나타내는 반면, (·)은 G-U 워블 염기쌍을 나타낸다. "가변성 20nt" 또는 "20nt"는 표적 DNA에 상보적인 DNA-표적화 세그먼트를 나타낸다(이 영역은 길이가 최대 약 100nt일 수 있다). 타겟터-RNA 및 활성제-RNA의 특징을 통합한 단일-분자 DNA-표적화 RNA의 설계가 또한 제시된다. (다양한 종으로부터 Cas9/Csn1 단백질 서열은 도 3에 묘사되고, 서열번호 1-256 및 795-1346으로서 나타난다) 스트렙토코쿠스 파이오제네스: 위에서 아래로: (서열번호 //, //, //); 리스테리아 이노쿠아: 위에서 아래로: (서열번호 //, //, //). 제공된 서열은 비-제한적인 예이고, 단일-분자 DNA-표적화 RNA 및 2-분자 DNA-표적화 RNA가 다양한 종으로부터 천연적으로 현존하는 서열을 기반으로 하여 설계될 수 있는 방법을 설명하기 위해 의도된다. 다양한 종으로부터 적합한 서열의 다양한 예는 하기와 같이 나타난다(Cas9 단백질: 서열번호 1-259; tracrRNAs: 서열번호 431-562, 또는 그의 보체; crRNAs: 서열번호 563-679, 또는 그의 보체; 및 단일-분자 DNA-표적화 RNA의 예: 서열번호 680-682).
도 10a-e는 Cas9가 2개의 RNA 분자에 의해 유도된 DNA 엔도뉴클레아제이다는 것을 도시한다. 도 E(위에서 아래로, 서열번호 278-280, 및 //).
도 11a-b는 Cas9가 표적 DNA에서 두 가닥을 절단하기 위해 두 뉴클레아제 도메인을 사용한다는 것을 입증한다.
도 12a-e는 표적 DNA의 Cas9-촉매된 절단이 tracrRNA에서 활성화 도메인을 필요로 하고, crRNA에서 씨드 서열에 의해 조절된다는 것을 설명한다. 도 12c (위에서 아래로, 서열번호 278-280, 및 //); 도 12D (위에서 아래로, 서열번호 281-290); 및 도 12e (위에서 아래로, 서열번호 291-292, 283, 293-298).
도 13a-c는 PAM이 Cas9-tracrRNA:crRNA 복합체에 의한 표적 DNA 절단을 허가하기 위해 필요하다는 것을 도시한다.
도 14a-c는 Cas9가 tracrRNA 및 crRNA 특징을 결합하는 단일 조작된 RNA 분자를 사용하여 프로그래밍될 수 있다는 것을 설명한다. 키메라 A (서열번호 299); 키메라 B (서열번호 300).
도 15는 유형 II RNA-매개된 CRISPR/Cas 면역 경로를 묘사한다.
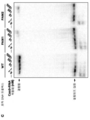
도 16a-b는 Cas9 뉴클레아제의 정제를 묘사한다.
도 17a-c는 이중-tracrRNA:crRNA에 의해 유도된 Cas9가 프로토스페이서 플라스미드 및 올리고뉴클레오타이드 DNA를 절단한다는 것을 도시한다. 도 17b (위에서 아래로, 서열번호 301-303, 및 //); 및 도 17c (위에서 아래로, 서열번호 304-306, 및 //).
도 18a-b는 Cas9가 3'-5' 엑소뉴클레아제 활성을 갖는 Mg2+-의존적 엔도뉴클레아제이다는 것을 도시한다.
도 19a-c는 표적 DNA의 이중-tracrRNA:crRNA 유도된 Cas9 절단이 부위 특이적이다는 것을 설명한다. 도 19c(위에서 아래로, 서열번호 307-309, //, 337-339, 및 //).
도 20a-b는 표적 DNA의 이중-tracrRNA:crRNA 유도된 Cas9 절단이 신속하고 효율적이다는 것을 도시한다.
도 21a-b는 Cas9의 HNH 및 RuvC-형 도메인이 각각 상보적 및 비상보적 DNA 가닥의 절단을 지시한다는 것을 도시한다.
도 22는 tracrRNA가 표적 DNA 인식에 필요하다는 것을 입증한다.
도 23a-b는 tracrRNA의 최소 영역은 표적 DNA의 이중-tracrRNA:crRNA 지시된 절단을 유도할 수 있다는 것을 도시한다.
도 24a-d는 Cas9에 의한 이중-tracrRNA:crRNA 유도된 표적 DNA 절단이 종 특이적일 수 있음을 입증한다.
도 25a-c는 crRNA에서 씨드 서열이 Cas9에 의한 표적 DNA의 이중 tracrRNA:crRNA 유도된 절단을 제어한다는 것을 도시한다. 도 25a: 표적 DNA 프로브 1 (서열번호 310); 스페이서 4 crRNA (1-42) (서열번호 311); tracrRNA (15-89) (서열번호 //). 도 25b 좌측 패널 (서열번호 310).
도 26a-c는 PAM 서열이 Cas9-tracrRNA:crRNA에 의한 프로토스페이서 플라스미드 DNA 절단 및 박테리어 세포에서 Cas9-매개된 플라스미드 DNA 간섭에 필수적임을 입증합다. 도 26b(위에서 아래로, 서열번호 312-314); 및 도 26c(위에서 아래로, 서열번호 315-320).
도 27a-c는 이중 tracrRNA:crRNA를 모방하는 단일 키메라 RNA에 의해 유도된 Cas9가 프로토스페이서 DNA를 절단함을 도시한다. 도 27c(위에서 아래로, 서열번호 321-324).
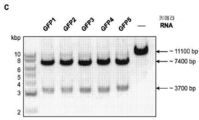
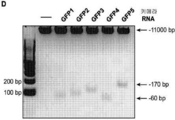
도 28a-d는 그린 형광 단백질 (GFP) 유전자 서열을 표적화하는 키메라 RNA의 드 노보 설계를 묘사한다. 도 28b(위에서 아래로, 서열번호 325-326). 도 28c: GFP1 표적 서열 (서열번호 327); GFP2 표적 서열 (서열번호 328); GFP3 표적 서열 (서열번호 329); GFP4 표적 서열 (서열번호 330); GFP5 표적 서열 (서열번호 331); GFP1 키메라 RNA (서열번호 332); GFP2 키메라 RNA (서열번호 333); GFP3 키메라 RNA (서열번호 334); GFP4 키메라 RNA (서열번호 335); GFP5 키메라 RNA (서열번호 336).
도 29a-e는 인간 세포에서 Cas9 및 가이드 RNA의 공-발현이 표적 유전자 좌에서 이중-가닥 DNA 파괴를 생성함을 입증한다. 도 29c(위에서 아래로, 서열번호 425-428).
도 30a-b는 세포 용해물이 활성 Cas9:sgRNA를 함유하고, 부위 특이적인 DNA 절단을 지지함을 입증한다.
도 31a-b는 sgRNA 작제물의 3' 확대가 부위 특이적인 NHEJ-매개된 돌연변이유발을 강화시킴을 입증한다. 도 31a(위에서 아래로, 서열번호 428-430).
도 32a-b는 다양한 유기체(A)로부터 대표적인 Cas9 서열의 계통수 뿐만 아니라 나무(B)의 주요 그룹에 대한 Cas9 유전자 좌위 구조를 묘사한다.
도 33a-e는 선택된 박테리아 종으로부터 유형 II CRISPR-Cas의 구조를 묘사한다.
도 34a-b는 선택된 유형 II CRISPR Cas 시스템에서 tracrRNA 및 전-crRNA 공-처리를 묘사한다. 도 34a(위에서 아래로, 서열번호 //,//,//,//,//,//,//,//); 도 34b(위에서 아래로, 서열번호 //,//,//,//).
도 35는 tracrRNA 서열의 다양성을 입증하는 tracrRNA 오쏘로그의 서열 정렬을 묘사한다.
도 36a-f는 딥 RNA 서열분석에 의해 나타낸 박테리아 tracrRNA 오쏘로그 및 crRNA의 발현을 묘사한다.
도 37a-o는 좌표(관심 영역) 및 상응하는 cDNA 서열(5' 내지 3')을 포함하여 연구된 박테리아 종에 대한 서열분석으로 검색된 모든 tracrRNA 오쏘로그 및 성숙한 crRNA를 나열한다.
도 38a-b는 서명 유전자 cas9의 존재를 특징으로 하는 유형 II CRISPR-Cas 유전자 좌를 함유하는 박테리아 종의 표를 제시한다. 이러한 서열은 계통발생적 분석에 사용되었다.
도 39a-b는 CRISPR 간섭(CRISPRi) 시스템의 설계를 묘사한다.
도 40a-e는 CRISPRi가 전사 스트레치 및 개시를 효과적으로 침묵시킴을 입증한다.
도 41a-b는 CRISPRi가 전사 스트레치를 차단함으로써 기능함을 입증한다.
도 42a-c는 CRISPRi 시스템의 표적화 특이성을 입증한다.
도 43a-f는 사일런싱 효율에 영향을 미치는 인자의 특성규명을 묘사한다.
도 44a-c는 CRISPRi 유전자 녹다운을 사용하는 복합체 조절 네트워크의 기능성 프로파일링을 묘사한다.
도 45a-b는 포유동물 세포에서 CRISPRi를 사용하는 유전자 사일런싱을 입증한다.
도 46은 에스. 파이오제네스로부터 유형 II CRISPR 시스템의 기전을 묘사한다.
도 47a-b는 dCas9 및 sgRNA로 공-형질감염된 이. 콜라이( coli ) 세포 배양물의 성장 곡선을 묘사한다.
도 48은 CRISPRi가 멀티-카피 플라스미드 상에서 리포터 유전자의 발현을 침묵시킬 수 있음을 도시한다.
도 49 A-C는 상이한 유전자를 표적화하는 sgRNA를 갖는 세포의 RNA-seq 데이타를 묘사한다.
도 50 A-E는 인접한 이중 불일치에 의한 sgRNA의 사일런싱 효과를 묘사한다.
도 51 A-C는 단일 유전자를 조절하기 위해 두 개의 sgRNA를 사용하는 조합 사일런싱 효과를 묘사한다.
도 52는 sgRNA 억제가 표적 유전자 좌 및 전사 개시로부터의 상대적 거리에 의존적이다는 것을 도시한다.
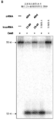
도 53 a-c는 변이체 Cas9 부위 지향적 폴리펩타이드(dCas9)가, dCas9가 유일한 RuvC1 도메인(예를 들면, D10A), 유일한 HNH 도메인(예를 들면, H840A), 또는 두 도메인(예를 들면, D10A 및 H840A)에서 감소된 활성을 가질 경우 당해 방법으로 작동함을 입증하는 실험 결과를 묘사한다.
도 54a-c는 당해 변이체 Cas9 부위 지향적 폴리펩타이드에 적당한 융합 파트너(또는 그의 단편)의 예들을 열거한다. 예들은 열거된 것들을 포함하지만, 이에 국한되지 않는다.
도 55a-d는 키메라 부위 지향적 폴리펩타이드가 인간 세포에서 전사를 활성화(증가)시키기 위해 사용될 수 있음을 입증한다.
도 56은 키메라 부위 지향적 폴리펩타이드가 인간 세포에서 전사를 억제(감소)하기 위해 사용될 수 있음을 입증한다.
도 57a-b는 자연 발생 tracrRNAs 및 crRNAs와 거의 50% 동일성을 공유하는 인공 서열이 DNA-표적화 RNA의 단백질-결합 도메인의 구조가 보존되는 한, 표적 DNA를 절단하기 위해 Cas9로 기능할 수 있음을 입증한다.
정의- 파트 I
본원에 상호교환적으로 사용된 용어 "폴리뉴클레오타이드" 및 "핵산"은 임의의 길이의 뉴클레이타이드의 중합체 형태, 리보뉴클레오타이드 또는 데옥시리보뉴클레오타이드를 의미한다. 따라서, 이 용어는 단일-, 이중- 또는 다중-가닥 DNA 또는 RNA, 게놈 DNA, cDNA, DNA-RNA 하이브리드, 또는 퓨린 및 피리미딘 염기 도는 다른 천연, 화학적으로 또는 생화학적으로 변형된 비천연, 또는 유도된 뉴클레오타이드 염기를 포함하는 중합체를 포함하지만, 이에 국한되지 않는다. "올리고뉴클레오타이드"는 일반적으로 단일- 또는 이중-가닥 DNA의 약 5 내지 약 100 개의 뉴클레오타이드의 폴리뉴클레오타이드를 의미한다. 그러나, 본 명세서의 목적을 위해, 올리고뉴클레오타이드의 길이에 대한 상한치는 없다. 올리고뉴클레오타이드는 또한 "올리고머" 또는 "올리고"로서 공지되어 있고, 유전자로부터 단리되거나 당해 기술 분야에 공지된 방법에 의해 화학적으로 합성될 수 있다. 용어 "폴리뉴클레오타이드" 및 "핵산"은 기재된 구현예에 적용가능한 것으로서, 단일-가닥(예: 센스 또는 안티센스) 및 이중-가닥 폴리뉴클레오타이드를 포함하는 것으로서 이해되어야 한다.
"줄기-루프 구조"는 주로 단일-가닥 뉴클레오타이드의 영역(루프부)에 의해 한쪽에 연결된 이중 가닥을 형성하는 것으로 공지되거나 예상된 뉴클레오타이드의 영역(단계부)을 포함하는 2차 구조를 갖는 핵산을 의미한다. 용어 "헤어핀" 및 "폴드-백" 구조는 또한 줄기-루프 구조를 의미하기 위해 본원에 사용된다. 이러한 구조물들은 당해 기술 분야에 익히 공지되어 있고, 이러한 용어들은 당해 기술 분야에 이들의 공지된 의미로 일관되게 사용된다. 당해 기술 분야에 공지된 바와 같이, 줄기-루프 구조는 정확한 염기 짝짓기를 필요로 하지 않는다. 따라서, 상기 줄기는 하나 이상의 염기 불일치를 포함할 수 있다. 대안적으로, 염기 짝짓기는 정확할 수 있다, 즉, 임의의 불일치를 포함하지 않을 수 있다.
"하이브리드화가능한" 또는 "상보적" 또는 "실질적으로 상보적"이란, 핵산(예: RNA)이 그것이 비공유적으로 결합하도록 하는, 즉, 왓슨-크릭 염기쌍 및/또는 G/U 염기쌍을 형성하고 온도 및 용액 이온 강도의 적절한 시험관내 및/또는 생체내 조건하에 서열 특이적 역평행 방식으로 또 하나의 핵산에 "어닐링하고" 또는 "하이브리드화하는"(즉, 핵산은 구체적으로 상보적 핵산에 결합한다) 뉴클레오타이드의 서열을 포함한다. 당해 기술 분야에 공지된 바와 같이, 표준 왓슨-크릭 염기 짝짓기는 아데닌(A)과 티미딘(T)의 짝짓기, 아데닌(A)과 우라실(U)의 짝짓기 및 구아닌(G)과 시토신(C)의 짝짓기[DNA, RNA]를 포함한다. 또한, 두 RNA 분자(예: dsRNA) 사이의 하이브리드화를 위해, 구아닌(G)과 우라실(U) 염기쌍이 당해 기술 분야에 공지되어 있다. 예를 들어, G/U 염기 짝짓기는 부분적으로 mRNA 중의 코돈과 tRNA 항-코돈 염기 짝짓기의 관점에서 유전자 코드의 축퇴(즉, 과잉)의 원인이다. 이 개시내용의 문맥에서, 당해 DNA-표적화 RNA 분자의 단백질 결합 세그먼트(dsRNA 듀플렉스)의 구아닌(G)은 우라실(U)에 상보적인 것으로 간주되고, 그 반대로 마찬가지이다. 이와 같이, G/U 염기쌍이 주어진 뉴클레오타이드 위치에서 당해 DNA 표적화 RNA 분자의 단백질 결합 세그먼트(dsRNA 듀플렉스)를 제조할 수 있을 경우, 그 위치는 비상보적인 것으로 간주되지 않고, 대신 상보적인 것으로 간주된다.
하이브리드화 및 세척 조건은 익히 공지되어 있고, 문헌[참조: Sambrook, J., Fritsch, E. F. and Maniatis, T. Molecular Cloning: A Laboratory Manual, Second Edition, Cold Spring Harbor Laboratory Press, Cold Spring Harbor (1989), particularly Chapter 11 and Table 11.1 therein; 및 Sambrook, J. and Russell, W., Molecular Cloning: A Laboratory Manual, Third Edition, Cold Spring Harbor Laboratory Press, Cold Spring Harbor (2001)]에 예시되어 있다. 온도 및 이온 강도의 조건은 하이브리드화의 "엄중함"을 결정한다.
하이브리드화는 염기 사이에 불일치가 가능하지만, 두 핵산이 상보적 서열을 함유할 것을 필요로 한다. 두 핵산 사이의 하이브리드화에 적합한 조건은 당해 기술 분야에 익히 공지된 변수인 핵산의 길이 및 상보성의 정도에 좌우된다. 두 뉴클레오타이드 서열 사이의 상보성의 정도가 클수록, 이러한 서열을 갖는 핵산의 하이브리드에 대한 융점(Tm) 값이 커진다. 상보성의 짧은 스트레치를 갖는 핵산(예: 상보성 35 이하, 30 이하, 25 이하, 22 이하, 20 이하 또는 18 개 이하의 뉴클레오타이드)의 하이브리드화를 위해, 불일치의 위치가 중요해진다(참조: Sambrook 등, 상기와 동일, 11.7-11.8). 전형적으로, 하이브리드화가능한 핵산의 길이는 적어도 약 10 개의 뉴클레오타이드이다. 하이브리드화가능한 핵산의 예시적인 최소 길이는 다음과 같다: 적어도 약 15 개의 뉴클레오타이드; 적어도 약 20 개의 뉴클레오타이드; 적어도 약 22 뉴클레오타이드; 적어도 약 25 개의 뉴클레오타이드 및 적어도 약 30 개의 뉴클레오타이드). 또한, 숙련가는 온도 및 세척 용액 염 농도가 필요에 따라 상보성 영역의 길이 및 상보성의 정도와 같은 인자에 따라 조정될 수 있음을 인지한다.
폴리뉴클레오타이드의 서열이 특이적으로 하이브리드화가능한 또는 하이브리드화가능한 이의 표적 핵산의 서열에 100% 상보적일 필요는 없다는 것이 당해 기술 분야에서 이해된다. 또한, 폴리뉴클레오타이드는 하나 이상의 세그먼트에 대해 하이브리화되어 개재 또는 인접한 세그먼트가 하이브리드화 이벤트(예: 루프 구조 또는 헤어핀 구조)에 연루되지 않도록 한다. 폴리뉴클레오타이드는 그들이 표적화되는 표적 핵산 서열 내의 표적 영역에 대해 적어도 70%, 적어도 80%, 적어도 90%, 적어도 95%, 적어도 99% 또는 100% 서열 상보성을 포함할 수 있다. 예를 들면, 안티센스 화합물의 18 내지 20 개의 뉴클레오타이드가 표적 영역에 대해 상보적이고 따라서 특이적으로 하이브리드화하는 안티센스 핵산은 90% 상보성을 나타낼 것이다. 이 예에서, 잔류하는 비상보적 뉴클레오타이드는 상보적 뉴클레오타이드와 군집형태화되거나 사이에 배치될 수 있고, 서로 또는 상보적 뉴클레오타이드와 인접할 필요는 없다. 핵산 내의 핵산 서열의 특별한 스트레치 사이의 상보성 퍼센트는 당해 기술 분야에 공지된 BLAST 프로그램(기초적인 국소 정렬 검색 도구) 및 PowerBLAST 프로그램(참조: Altschul 등, J. Mol. Biol., 1990, 215, 403-410; Zhang and Madden, Genome Res., 1997, 7, 649-656)을 사용하여 또는 갭 프로그램(참조: Wisconsin Sequence Analysis Package, Version 8 for Unix, Genetics Computer Group, University Research Park, Madison Wis.)을 사용하여, 스미스(Smith) 및 워터만(Waterman)의 알고리즘을 사용하는 디폴트 셋팅(참조: Adv. Appl. Math., 1981, 2, 482-489)을 사용하여 일상적으로 결정될 수 있다.
용어 "펩타이드", "폴리펩타이드" 및 "단백질"은 본원에서 상호교환적으로 사용되고, 임의의 길이의 아미노산의 중합체 형태를 의미하고, 이는 코드화 및 비코드화 아미노산, 화학적으로 또는 생화학적으로 변형되거나 유도된 아미노산, 및 변형된 펩타이드 골격을 갖는 폴리펩타이드를 포함할 수 있다.
본원에서 사용된 "결합"(예: 폴리펩타이드의 RNA-결합 도메인을 참조)은 거대분자 사이(예: 단백질과 핵산 사이)의 비공유 상호작용을 의미한다. 비공유 상호작용의 상태에서, 거대분자는 "연관된" 또는 "상호작용" 또는 "결합"하는 것으로 간주된다(예를 들어, 분자 X가 분자 Y와 상호작용하는 것으로 간주되면, 이는 분자 X가 비공유 방식으로 분자 Y에 결합한다는 것을 의미한다). 결합 상호작용의 모든 성분이 서열 특이적일(예: DNA 골격 중의 포스페이트 잔기와 접촉할) 필요는 없지만, 결합 상호작용의 몇몇 부분은 서열 특이적일 수 있다. 결합 상호작용은 일반적으로 10-6M 미만, 10-7M 미만, 10-8M 미만, 10-9M 미만, 10-10M 미만, 10-11M 미만, 10-12M 미만, 10-13M 미만, 10-14M 미만 또는 10-15M 미만의 해리 상수(Kd)를 특징으로 한다. "친화성"은 결합 강도를 의미하고, 증가된 결합 친화성은 낮은 Kd와 상관된다.
"결합 도메인"이란, 또 하나의 분자에 비공유적으로 결합할 수 있는 단백질 도메인을 의미한다. 결합 도메인은, 예를 들면, DNA 분자(DNA-결합 단백질), RNA 분자(RNA-결합 단백질) 및/또는 단백질 분자(단백질-결합 단백질)에 결합할 수 있다. 단백질 도메인-결합 단백질의 경우에, 이는 (동종이량체, 동종삼량체 등을 형성하기 위해) 그 자체에 결합할 수 있고/있거나 상이한 단백질 또는 단백질들의 하나 이상의 분자에 결합할 수 있다.
용어 "보존적 아미노산 치환"은 유사한 측쇄를 갖는 아미노산 잔기의 단백질에서 교환성을 의미한다. 예를 들면, 지방족 측쇄를 갖는 아미노산의 그룹은 글리신, 알라닌, 발린, 류신 및 이소류신으로 이루어지고; 지방족 하이드록실 측쇄를 갖는 아미노산의 그룹은 세린 및 트레오닌으로 이루어지고; 아미드 함유 측쇄를 갖는 아미노산의 그룹은 아스파라긴 및 글루타민으로 이루어지고; 방향족 측쇄를 갖는 아미노산의 그룹은 페닐알라닌, 티로신 및 트립토판으로 이루어지고; 염기성 측쇄를 갖는 아미노산의 그룹은 라이신, 아르기닌 및 히스티딘으로 이루어지고; 산성 측쇄를 갖는 아미노산의 그룹은 글루타메이트 및 아스파르테이트로 이루어지고; 황 함유 측쇄를 갖는 아미노산의 그룹은 시스테인 및 메티오닌으로 이루어진다. 예시적인 보존적 아미노산 치환 그룹은 발린-류신-이소류신, 페닐알라닌-티로신, 라이신-아르기닌, 알라닌-발린 및 아스파라긴-글루타민이다.
폴리뉴클로타이드 또는 폴리펩타이드는 또 하나의 폴리뉴클레오타이드 또는 폴리펩타이드와 특정 퍼센트의 "서열 동일성"을 갖고, 정렬될 경우, 그 퍼센트의 염기 또는 아미노산이 동일하고, 두 서열을 비교할 경우 동일한 대응 위치에 존재한다는 것을 의미한다. 서열 동일성은 수많은 상이한 방식으로 결정될 수 있다. 서열 동일성을 결정하기 위해, 서열은 ncbi.nlm.nili.gov/BLAST, ebi.ac.uk/Tools/msa/tcoffee/, ebi.ac.uk/Tools/msa/muscle/, mafft.cbrc.jp/alignment/software/를 포함하는 전 세계적인 웹 사이트에서 이용가능한 다양한 방법 및 컴퓨터 프로그램(예: BLAST, T-COFFEE, MUSCLE, MAFFT 등)을 사용하여 정렬시킬 수 있다(참조: 예를 들어, Altschul 등 (1990), J. Mol. Bioi. 215:403-10).
특별한 RNA를 "인코딩"하는 DNA 서열은 RNA로 전사되는 DNA 핵산 서열이다. DNA 폴리뉴클레오타이드는 단백질로 번역되는 RNA(mRNA)를 인코딩할 수 있거나, DNA 폴리뉴클레오타이드는 단백질로 번역되지 않은 RNA(예: tRNA, rRNA, 또는 DNA-표적화 RNA; 또한 "비코딩" RNA 또는 "ncRNA"라 칭명됨)를 인코딩할 수 있다.
"단백질 코딩 서열" 또는 특별한 단백질 또는 폴리펩타이드를 인코딩하는 서열은 mRNA(DNA의 경우)로 전사되고, 적합한 조절 서열의 조절하에 위치시킬 때, 시험관내 또는 생체내에서 폴리펩타이드로 번역되는(mRNA의 경우) 핵산 서열이다. 코팅 서열의 경계는 5' 말단(N-말단)에서 시작 코돈 및 3' 말단(C-말단)에서 번역 정지 논센스 코돈에 의해 결정된다. 코딩 서열은 원핵 또는 진핵 mRNA으로부터의 cDNA, 원핵 또는 진핵 DNA로부터의 게놈 DNA 서열 및 합성 핵산을 포함할 수 있지만, 이에 국한되지 않는다. 전사 종결 서열은 일반적으로 코팅 서열에서 3'에 위치될 것이다.
본원에 사용된 "프로모터 서열"은 RNA 폴리머라제를 결합시키고 다운스트림(3' 방향) 코딩 또는 비-코딩 서열의 전사를 개시할 수 있는 DNA 조절 영역이다. 본 발명을 정의하기 위해, 프로모터 서열은 전사 개시 부위에 의해 이의 3' 말단에서 결합되고 배경 이상의 검출가능한 수준에서 전사를 개시하는데 필요한 염기 또는 요소의 최소 수를 포함하기 위해 업스트림(5' 방향) 연장된다. 프로모터 서열 내에서, 전사 개시 부위 뿐만 아니라 RNA 폴리머라제의 결합의 원인인 단백질 결합 도메인이 발견될 것이다. 진핵 프로모터는 항상은 아니지만 자주 "TATA" 박스 및 "CAT" 박스를 함유한다. 유도성 프로모터를 포함하는 다양한 프로모터를 사용하여 본 발명의 다양한 벡터를 구동할 수 있다.
프로모터는 구조적으로 활성 프로모터(즉, 구조적으로 활성/"온" 상태로 존재하는 프로모터)일 수 있고, 이는 유도성 프로모터(즉, 상태 활성/"온" 또는 불활성/"오프"가 외부 자극, 예를 들면, 특정한 온도, 화합물 또는 단백질의 존재로 조절되는 프로모터)일 수 있고, 이는 공간적으로 제한된 프로모터(즉, 전사 조절 요소, 인핸서 등)(예: 조직 특이적 프로모터, 세포형 특이적 프로모터 등)일 수 있고, 이는 일시적으로 제한된 프로모터일 수 있다(즉, 프로모터는 배아 발생의 특이적 단계 동안 또는 생물학적 과정의 특이적 단계 동안, 예를 들어, 마우스의 모낭 사이클 동안 "온" 상태 또는 "오프" 상태로 존재한다).
적합한 프로모터는 바이러스로부터 유도될 수 있고, 따라서 바이러스 프로모터로서 칭명될 수 있거나, 그들은 원핵 또는 진핵 유기체를 포함하는 임의의 유기체로부터 유도될 수 있다. 적합한 프로모터를 사용하여 임의의 RNA 폴리머라제(예: pol I, pol II, pol III)에 의한 발현을 구동시킬 수 있다. 예시적인 프로모터는 SV40 초기 프로모터, 마우스 유선 종양 바이러스 긴 말단 반복(LTR) 프로모터, 아데노바이러스 주요 후기 프로모터(Ad MLP); 단순 포진 바이러스(HSV) 프로모터, 사이토메갈로바이러스(CMV) 프로모터, 예를 들면, CMV 전초기 프로모터 영역(CMVIE), 라우스 육종 바이러스(RSV) 프로모터, 인간 U6 소핵 프로모터(U6)(예: Miyagishi 등 , Nature Biotechnology 20, 497 - 500 (2002)), 증대된 U6 프로모터(예: Xia 등, Nucleic Acids Res. 2003 Sep 1;31(17)), 인간 H1 프로모터(H1) 등을 포함하지만, 이에 국한되지 않는다.
유도성 프로모터의 예는 T7 RNA 폴리머라제 프로모터, T3 RNA 폴리머라제 프로모터, 이소프로필-베타-D-티오갈락토피라노사이드(IPTG)-조절된 프로모터, 락토오스 유도된 프로모터, 열 충격 프로모터, 테트라사이클린-조절된 프로모터, 스테로이드-조절된 프로모터, 금속-조절된 프로모터, 에스트로겐 수용체-조절된 프로모터 등을 포함하지만, 이에 국한되지 않는다. 따라서, 유도성 프로모터는 독시사이클린; RNA 폴리머라제, 예를 들면, T7 RNA 폴리머라제; 에스트로겐 수용체; 에스트로겐 수용체 융합 등을 포함하지만, 이에 국한되지 않는 분자로 조절될 수 있다.
일부 구현예에서, 프로모터는 다중-세포성 유기체에서, 프로모터가 특이적 세포의 서브 세트에서 활성(즉, "온")이도록 하는 공간적으로 제한된 프로모터(즉, 세포형 특이적 프로모터, 조직 특이적 프로모터 등)이다. 공간적으로 제한된 프로모터는 또한 인핸서, 전사 조절 요소, 제어 서열 등으로서 칭명될 수 있다. 임의의 편리한 공간적으로 제한된 프로모터가 사용될 수 있고, 적합한 프로모터(예: 뇌 특이적 프로모터, 뉴런의 서브세트에서 발현을 구동하는 프로모터, 생식계열에서 발현을 구동하는 프로모터, 폐에서 발현을 구동하는 프로모터, 근육에서 발현을 구동하는 프로모터, 췌장의 소도세포에서 발현을 구동하는 프로모터 등)의 선택은 유기체에 좌우된다. 예를 들면, 다양한 공간적으로 제한된 프로모터는 식물, 나는 것, 벌레, 포유동물, 마우스 등에 대해 공지되어 있다. 따라서, 공간적으로 제한된 프로모터는 유기체에 따라 각종 다양한 상이한 조직 및 세포 형태에서 당해 부위 지향적 변형 폴리펩타이드를 인코딩하는 핵산의 발현을 조절하기 위해 사용될 수 있다. 일부 공간적으로 제한된 프로모터는 또한 프로모터가 배아 발생의 특이적 단계 동안 또는 생물학적 과정의 특이적 단계 동안(예: 마우스의 모낭 사이클) "온" 상태 또는 "오프" 상태로 존재하도록 일시적으로 제한된다.
설명하기 위해, 공간적으로 제한된 프로모터의 예는 뉴런-특이적 프로모터, 지방세포-특이적 프로모터, 심근세포-특이적 프로모터, 평활근-특이적 프로모터, 광수용체-특이적 프로모터 등을 포함하지만, 이에 국한되지 않는다. 뉴런-특이적 공간적으로 제한된 프로모터는 뉴런-특이적 에놀라제(NSE) 프로모터(참조: 예를 들면, EMBL HSENO2, X51956); 방향족 아미노산 데카복실라제(AADC) 프로모터; 신경필라멘트 프로모터(참조: 예를 들면, GenBank HUMNFL, L04147); 시냅신 프로모터(참조: 예를 들면, GenBank HUMSYNIB, M55301); thy-1 프로모터(참조: 예를 들면, Chen 등 (1987) Cell 51:7-19; 및 Llewellyn, 등 (2010) Nat. Med. 16(10):1161-1166); 세로토닌 수용체 프로모터(참조: 예를 들면, GenBank S62283); 티로신 하이드록실라제 프로모터(TH)(참조: 예를 들면, Oh 등 (2009) Gene Ther 16:437; Sasaoka 등 (1992) Mol. Brain Res. 16:274; Boundy 등 (1998) J. Neurosci. 18:9989; 및 Kaneda 등 (1991) Neuron 6:583-594); GnRH 프로모터(참조: 예를 들면, Radovick 등 (1991) Proc. Natl. Acad. Sci. USA 88:3402-3406); L7 프로모터(참조: 예를 들면, Oberdick 등 (1990) Science 248:223-226); DNMT 프로모터(참조: 예를 들면, Bartge 등 (1988) Proc. Natl. Acad. Sci. USA 85:3648-3652); 엔케팔린 프로모터(참조: 예를 들면, Comb 등 (1988) EMBO J. 17:3793-3805); 수초 염기성 단백질(MBP) 프로모터; Ca2+-칼모둘린-의존적 단백질 키나제 II-알파(CamKIIα) 프로모터(참조: 예를 들면, Mayford 등 (1996) Proc. Natl. Acad. Sci. USA 93:13250; 및 Casanova 등 (2001) Genesis 31:37); CMV 인핸서/혈소판-유도된 성장 인자-β 프로모터(참조: 예를 들면, Liu 등 (2004) Gene Therapy 11:52-60) 등을 포함하지만, 이에 국한되지 않는다.
지방세포-특이적 공간적으로 제한된 프로모터는 aP2 유전자 프로모터/인핸서, 예를 들면, 인간 aP2 유전자의 -5.4kb 내지 +21bp의 영역(참조: 예를 들면, Tozzo 등 (1997) Endocrinol. 138:1604; Ross 등 (1990) Proc. Natl. Acad. Sci. USA 87:9590; 및 Pavjani 등 (2005) Nat. Med. 11:797); 글루코스 수송체-4(GLUT4) 프로모터(참조: 예를 들면, Knight 등 (2003) Proc. Natl. Acad. Sci. USA 100:14725); 지방산 트랜스로카제 (FAT/CD36) 프로모터(참조: 예를 들면, Kuriki 등 (2002) Biol. Pharm. Bull. 25:1476; 및 Sato 등 (2002) J. Biol. Chem. 277:15703); 스테아로일-CoA 데사투라제-1 (SCD1) 프로모터(참조: Tabor 등 (1999) J. Biol. Chem. 274:20603); 렙틴 프로모터(참조: 예를 들면, Mason 등 (1998) Endocrinol. 139:1013; 및 Chen 등 (1999) Biochem. Biophys. Res. Comm. 262:187); 아디포넥틴 프로모터(참조: 예를 들면, Kita 등 (2005) Biochem. Biophys. Res. Comm. 331:484; 및 Chakrabarti (2010) Endocrinol. 151:2408); 아딥신 프로모터(참조: 예를 들면, Platt 등 (1989) Proc. Natl. Acad. Sci. USA 86:7490); 레지스틴 프로모터 (참조: 예를 들면, Seo 등 (2003) Molec. Endocrinol. 17:1522) 등을 포함하지만, 이에 국한되지 않는다.
심근세포-특이적 공간적으로 제한된 프로모터는 다음 유전자로부터 유도된 제어 서열을 포함하지만, 이에 국한되지 않는다: 미오신 경쇄-2, α-미오신 중쇄, AE3, 심장 트로포닌 C, 심장 액틴 등(참조: Franz 등 (1997) Cardiovasc. Res. 35:560-566; Robbins 등 (1995) Ann. N.Y. Acad. Sci. 752:492-505; Linn 등 (1995) Circ. Res. 76:584-591; Parmacek 등 (1994) Mol. Cell. Biol. 14:1870-1885; Hunter 등 (1993) Hypertension 22:608-617; 및 Sartorelli 등 (1992) Proc. Natl. Acad. Sci. USA 89:4047-4051).
평활근-특이적 공간적으로 제한된 프로모터는 SM22α 프로모터(참조: 예를 들면, 등 (2000) Mol. Med. 6:983; 및 미국 특허 제7,169,874호); 스무텔린 프로모터(참조: 예를 들면, WO 2001/018048); α-평활근 액틴 프로모터 등을 포함하지만, 이에 국한되지 않는다. 예를 들면, 두 CArG 요소가 존재하는 SM22α 프로모터의 0.4 kb 영역은 혈관 평활근 세포-특이적 발현을 매개하는 것으로 나타났다(참조: 예를 들면, Kim, 등 (1997) Mol. Cell. Biol. 17, 2266-2278; Li, 등, (1996) J. Cell Biol. 132, 849-859; 및 Moessler, 등 (1996) Development 122, 2415-2425).
광수용체-특이적 공간적으로 제한된 프로모터는 로돕신 프로모터; 로돕신 키나아제 프로모터(참조: Young 등 (2003) Ophthalmol. Vis. Sci. 44:4076); 베타 포스포디에스테라제 유전자 프로모터(참조: Nicoud 등 (2007) J. Gene Med. 9:1015); 색소성 망막염 유전자 프로모터(참조: Nicoud 등 (2007) 상기와 동일); 상호광수용체 레티노이드-결합 단백질(IRBP) 유전자 인핸서(참조: Nicoud 등 (2007) 상기와 동일); IRBP 유전자 프로모터(참조: Yokoyama 등 (1992) Exp Eye Res. 55:225) 등을 포함하지만, 이에 국한되지 않는다.
본원에서 상호교환적으로 사용된 용어 "DNA 조절 서열", "조절 요소" 및 "조절 요소"는 비-코딩 서열(예: DNA-표적화 RNA) 또는 코딩 서열(예: 부위 지향적 변형 폴리펩타이드 또는 Cas9/Csn1 폴리펩타이드)의 전사를 제공하고/하거나 조절하고/하거나 인코딩된 폴리펩타이드의 번역을 조절하는 전사 및 번역 조절 서열, 예를 들면, 프로모터, 인핸서, 폴리아데닐화 신호, 종결자, 단백질 분해 신호 등을 의미한다.
핵산, 폴리펩타이드, 세포 또는 유기체에 적용될 때 본원에 사용된 용어 "자연 발생" 또는 "비변형된"은 자연에서 발견되는 핵산, 폴리펩타이드, 세포 또는 유기체를 의미한다. 예를 들면, 자연의 공급원으로부터 단리될 수 있고, 실험실에서 인간에 의해 의도적으로 변형되지 않은 유기체(바이러스 포함)에 존재하는 폴리펩타이드 또는 폴리뉴클레오타이드 서열은 자연 발생적이다.
핵산 또는 폴리펩타이드에 적용될 때 본원에 사용된 용어 "키메라"는 상이한 공급원으로부터 유도된 구조로 규정된 2개의 성분을 의미한다. 예를 들면, "키메라"가 키메라 폴리펩타이드(예: 키메라 Cas9/Csn1 단백질)의 문맥에서 사용될 경우, 키메라 폴리펩타이드는 상이한 폴리펩타이드로부터 유도된 아미노산 서열을 포함한다. 키메라 폴리펩타이드는 변형되거나 자연 발생 폴리펩타이드 서열(예: 변형되거나 변형되지 않은 Cas9/Csn1 단백질로부터 제 1 아미노산 서열 및 Cas9/Csn1 단백질 이외의 제 2 아미노산 서열)을 포함할 수 있다. 유사하게, 키메파 폴리펩타이드를 인코딩하는 폴리뉴클레오타이드의 문맥에서 "키메라"는 상이한 코딩 영역으로부터 유도된 뉴클레오타이드 서열(예: 변형되거나 변형되지 않은 Cas9/Csn1 단백질을 인코딩하는 제 1 뉴클레오타이드 서열 및 Cas9/Csn1 단백질 이외의 폴리펩타이드를 인코딩하는 제 2 뉴클레오타이드 서열)을 포함한다.
용어 "키메라 폴리펩타이드"는 보통 인간 개입을 통해 아미노 서열의 두 개의 달리 분리된 세그먼트의 조합(즉, "융합")으로 제조된 폴리펩타이드를 의미한다. 키메라 아미노산 서열을 포함하는 폴리펩타이드는 키메라 폴리펩타이드이다. 일부 키메라 폴리펩타이드는 "융합 변이체"로서 칭명될 수 있다.
본원에 사용된 "이종기원"은 각각 천연 핵산 또는 단백질에서 발견되지 않는 뉴클레오타이드 또는 폴리펩타이드 서열을 의미한다. 예를 들어, 키메라 Cas9/Csn1 단백질에서, 자연 발생 박테리아 Cas9/Csn1 폴리펩타이드(또는 이의 변이체)의 RNA-결합 도메인을 이종기원 폴리펩타이드 서열(Cas9/Csn1 이외의 단백질로부터의 폴리펩타이드 서열 또는 또 하나의 유기체로부터의 폴리펩타이드 서열)에 융합시킬 수 있다. 이종기원 폴리펩타이드 서열은 키메라 Cas9/Csn1 단백질에 의해 또한 나타나는 활성(예: 효소 활성)(예: 메틸트랜스퍼라제 활성, 아세틸트랜스퍼라제 활성, 키나제 활성, 우비퀴틴화 활성 등)을 나타낼 수 있다. 이종기원 핵산 서열은 자연 발생 핵산 서열(또는 이의 변이체)(예: 유전공학에 의해)에 연결되어 키메라 폴리펩타이드를 인코딩하는 키메라 뉴클레오타이드 서열을 생성할 수 있다. 또 하나의 예로서, 융합 변이체 Cas9 부위 지향적 폴리펩타이드에서, 변이체 Cas9 부위 지향적 폴리펩타이드는 이종기원 폴리펩타이드(즉, Cas9 이외의 폴리펩타이드)에 융합시킬 수 있고, 이는 융합 변이체 Cas9 부위 지향적 폴리펩타이드에 의해 또한 나타나는 활성을 나타낸다. 이종기원 핵산 서열은 변이체 Cas9 부위 지향적 폴리펩타이드(예: 유전공학에 의해)에 연결되어 융합 변이체 Cas9 부위 지향적 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 생성할 수 있다.
본원에 사용된 "재조합"은 특정한 핵산(DNA 또는 RNA)이 자연계에서 발견되는 내인성 핵산으로부터 구별할 수 있는 구조적 코딩 또는 비-코딩 서열을 갖는 작제물을 유도하는 클로닝, 제한, 폴리머라제 연쇄 반응(PCR) 및/또는 결찰 단계의 다양한 조합의 생성물임을 의미한다. 폴리펩타이드를 인코딩하는 DNA 서열은 cDNA 단편으로부터 또는 일련의 합성 올리고뉴클레오타이드로부터 조립되어 세포 또는 무세포 전사 및 번역 시스템에 함유된 재조합 전사 유닛으로부터 발현될 수 있는 합성 핵산을 제공할 수 있다. 관련된 서열을 포함하는 게놈 DNA는 또한 재조합 유전자 또는 전사 유닛의 형성에 사용될 수 있다. 비-번역된 DNA의 서열은 개방 판독 프레임으로부터 5' 또는 3'에 존재할 수 있고, 여기서 이러한 서열은 코딩 영역의 조작 또는 발현을 간섭하지 않고, 사실상 다양한 기전(참조: 이하 "DNA 조절 서열")에 의해 목적하는 생성물의 생산을 조절하도록 작용할 수 있다. 대안적으로, 번역되지 않은 RNA를 인코딩하는 DNA 서열(예: DNA-표적화 RNA)은 또한 재조합형으로 간주될 수 있다. 따라서, 예를 들면, 용어 "재조합" 핵산은 자연 발생이 아닌, 예를 들면, 인간 개입을 통해 서열의 두 개의 달리 분리된 세그먼트의 인공 조합에 의해 제조된 것을 의미한다. 이 인공 조합은 흔히 화학적 합성 수단에 의해 또는 핵산의 단리된 세그먼트의 인공 조작에 의해, 예를 들면, 유전공학 기술에 의해 달성된다. 이러한 것은 보통 코돈을 동일한 아미노산, 보존적 아미노산 또는 비-보존적 아미노산을 인코딩하는 코돈으로 교환하기 위해 수행된다. 대안적으로, 이는 목적하는 기능의 조합을 생성하기 위해 목적하는 기능의 핵산 세그먼트를 함께 결합시키기 위해 수행된다. 이 인공 조합은 흔히 화학적 합성 수단에 의해, 또는 핵산의 단리된 세그먼트의 인공 조작에 의해, 예를 들면, 유전공학 기술에 의해 달성된다. 재조합 폴리뉴클레오타이드가 폴리펩타이드를 인코딩할 때, 인코딩된 폴리펩타이드의 서열은 자연 발생적("야생형")일 수 있거나, 자연 발생 서열의 변이체(예: 돌연변이체)일 수 있다. 따라서, 용어 "재조합" 폴리펩타이드는 반드시 서열이 자연적으로 발생하지 않은 폴리펩타이드를 의미하지는 않는다. 대신에, "제조합" 폴리펩타이드는 재조합 DNA 서열에 의해 인코딩되지만, 폴리펩타이드의 서열은 자연 발생적("야생형") 또는 비-자연 발생적(예: 변이체, 돌연변이체 등)일 수 있다. 따라서, "재조합" 폴리펩타이드는 인간 개입 결과이지만, 자연 발생 아미노산 서열일 수 있다.
"벡터" 또는 "발현 벡터"는 세포에서 부착된 세그먼트의 복제를 일으키기 위해 또 하나의 DNA 세그먼트, 즉, "삽입물"이 부착될 수 있는 레플리콘, 예를 들면, 플라스미드, 파아지, 바이러스 또는 코스미드이다.
"발현 카세트"는 프로모터에 작동가능하게 연결된 DNA 코딩 서열을 포함한다. "작동가능하게 연결된"은 그렇게 기재된 성분이 그들의 의도된 방식으로 그들이 기능하도록 허용하는 관계로 존재하는 병렬을 의미한다. 예를 들면, 프로모터는, 프로모터가 이의 전사 또는 발현에 영향을 미칠 경우, 코딩 서열에 작동가능하게 연결된다.
용어들 "재조합 발현 벡터" 또는 "DNA 작제물"는 벡터 및 적어도 하나의 삽입물을 포함하는 DNA 분자를 의미하기 위해 본원에서 상호교환적으로 사용된다. 재조합 발현 벡터는 일반적으로 삽입물(들)의 발현 및/또는 증식을 위해 또는 기타 재조합 뉴클레오타이드 서열의 구성을 위해 생성된다. 이 삽입물(들)은 프로모터 서열에 작동가능하게 연결될 수 있거나 연결되지 않을 수 있고, DNA 조절 서열에 작동가능하게 연결될 수 있거나 연결되지 않을 수 있다.
세포는, 외인성 DNA가 세포 내부에 도입될 경우에 외인성 DNA, 예를 들면, 재조합 발현 벡터에 의해 "유전적으로 변형되고" 또는 "형질전환되고" 또는 "형질감염된다". 외인성 DNA의 존재는 영구적이거나 일시적인 유전적 변화를 유도한다. 형질전환 DNA는 세포의 게놈에 통합(공유 결합)될 수 있거나 되지 않을 수 있다. 예를 들면, 원핵생물, 효모 및 포유동물 세포에서, 형질전환 DNA는 에피솜 요소, 예를 들면, 플라스미드 상에서 유지시킬 수 있다. 진핵 세포에 대하여, 안정하게 형질전환된 세포는 형질전환 DNA가 염색체에 통합되어 염색체 복제를 통해 딸 세포에 의해 유전되도록 하는 것이다. 이 능력은 형질전환 DNA를 함유하는 딸 세포의 모집단을 포함하는 세포주 또는 클론을 확립하는 진핵 세포의 능력에 의해 입증된다. "클론"은 유사분열에 의해 단일 세포 또는 공통 조상으로부터 유도된 세포의 모집단이다. "세포주"는 많은 세대에 대해 시험관내에서 안정한 성장을 가능하게 하는 1차 세포의 클론이다.
적합한 유전적 변형 방법(또한 "형질전환"으로 칭명됨)은, 예를 들면, 바이러스 또는 박테리오파지 감염, 형질감염, 접합, 원형질 융합, 리포펙션, 전기천공, 인산칼슘 침전, 폴리에틸렌이민(PEI)-매개된 형질감염, DEAE-덱스트란 매개된 형질감염, 리포솜-매개된 형질감염, 입자총 기술, 인산칼슘 침전, 직접 미세 주입, 나노입자-매개된 핵산 전달(참조: Panyam 등 Adv Drug Deliv Rev. 2012 Sep 13. pii: S0169-409X(12)00283-9. doi: 10.1016/j.addr.2012.09.023) 등을 포함한다.
유전적 변형 방법의 선택은 일반적으로 형질전환되는 세포의 형태 및 형질전환이 일어나는 환경(예: 시험관내, 생체외 또는 생체내)에 의존적이다. 이러한 방법의 일반적인 논의는 문헌(참조: Ausubel, 등, Short Protocols in Molecular Biology, 3rd ed., Wiley & Sons, 1995)에서 찾을 수 있다.
본원에 사용된 "표적 DNA"는 "표적 부위" 또는 "표적 서열"을 포함하는 DNA 폴리뉴클레오타이드이다. 용어들 "표적 부위" 또는 "표적 서열" 또는 "표적 프로토스페이서 DNA"는 당해 DNA-표적화 RNA의 DNA-표적화 세그먼트가 결합하는(참조: 도 1 및 도 39) 표적 DNA에 존재하는 핵산 서열을 의미하기 위해 본원에서 상호교환적으로 사용되고, 단 결합에 충분한 조건이 존재한다. 예를 들면, 표적 DNA 내의 표적 부위(또는 표적 서열) 5'-GAGCATATC-3' (서열번호 //)는 RNA 서열 5'-GAUAUGCUC-3'(서열번호 //) 에 의해 표적화된다(또는 이에 의해 결합되거나, 이에 의해 하이브리드화되거나 이에 상보적이다). 적합한 DNA/RNA 결합 조건은 세포에 정상적으로 존재하는 생리적 조건을 포함한다. 기타 적합한 DNA/RNA 결합 조건(예: 무세포 시스템에서의 조건)은 당해 기술 분야에 공지되어 있다(참조: Sambrook, 상기와 동일). DNA-표적화 RNA에 상보적이고 이에 의해 하이브리화되는 표적 DNA의 가닥은 "상보적 가닥"으로서 칭명되고, "상보적 가닥"에 상보적인 (따라서 DNA-표적화 RNA에 상보적이지 않은) 표적 DNA의 가닥은 "비상보적 가닥" 또는 비-상보적 가닥"으로서 칭명된다(참조: 도 12).
"부위 지향적 변형 폴리펩타이드" 또는 " RNA-결합 부위 지향적 폴리펩타이드" 또는 "RNA-결합 부위 지향적 변형 폴리펩타이드" 또는 "부위 지향적 폴리펩타이드"란, RNA를 결합하고, 특이적 DNA 서열에서 표적화되는 폴리펩타이드를 의미한다. 본원에서 기술된 바와 같은 부위 지향적 변형 폴리펩타이드는 그것이 결합되는 RNA 분자에 의해 특이적 DNA 서열에서 표적화된다. RNA 분자는 표적 DNA 내에서 표적 서열에 상보적인 서열을 포함하고, 따라서 표적 DNA(표적 서열) 내의 특이적 위치로 결합된 폴리펩타이드를 표적화한다.
"절단"이란, DNA 분자의 공유 골격의 파괴를 의미한다. 절단은 포스포디에스테르 결합의 효소적 또는 화학적 가수분해를 포함하지만, 이에 국한되지 않는 다양한 방법에 의해 개시될 수 있다. 단일-가닥 절단 및 이중-가닥 절단 둘 다 가능하고, 이중-가닥 절단은 두 개의 뚜렷이 다른 단일-가닥 절단 이벤트의 결과로서 일어날 수 있다. DNA 절단은 무딘 말단 또는 엇갈린 말단의 생산을 유도할 수 있다. 특정 구현예에서, DNA-표적화 RNA 및 부위 지향적 변형 폴리펩타이드를 포함하는 복합체를 표적화된 이중-가닥 DNA 절단용으로 사용한다.
"뉴클레아제" 및 "엔도뉴클레아제"는 DNA 절단을 위한 촉매적 활성을 포함하는 효소를 의미하기 위해 본원에서 상호교환적으로 사용된다.
뉴클레아제의 "절단 도메인" 또는 "활성 도메인" 또는 "뉴클레아제 도메인"이란, DNA 절단을 위한 촉매적 활성을 포함하는 뉴클레아제 내의 폴리펩타이드 서열 또는 도메인을 의미한다. 절단 도메인은 단일 폴리펩타이드 쇄에 함유될 수 있거나 절단 활성은 두 개(또는 그 이상)의 폴리펩타이드의 회합으로부터 생성될 수 있다. 단일 뉴클레아제 도메인은 주어진 폴리펩타이드 내의 아미노산의 하나 이상의 단리된 스트레치로 이루어질 수 있다.
부위 지향적 변형 폴리펩타이드에 결합하고 표적 DNA 내의 특이적 위치에서 폴리펩타이드를 표적화하는 RNA 분자는 본원에서 "RNA-표적화 RNA" 또는 "DNA-표적화 RNA 폴리뉴클레오티드"로서 칭명된다(또한, 본원에서 "가이드 RNA" 또는 "gRNA"로서 칭명된다). 당해 DNA-표적화 RNA는 두 개의 세그먼트, DNA-표적화 세그먼트" 및 "단백질 결합 세그먼트"을 포함한다. "세그먼트"란, 분자의 세그먼트/섹션/영역, 예를 들면, RNA 중의 뉴클레오타이드의 인접 스트레치를 의미한다. 세그먼트는 또한 세그먼트가 하나 이상의 분자의 영역을 포함할 수 있도록 하는 복합체의 영역/섹션을 의미할 수 있다. 예를 들면, 일부 경우에 DNA-표적화 RNA의 단백질-결합 세그먼트(이하 기술됨)은 하나의 RNA 분자이고, 따라서 단백질-결합 세그먼트는 그 RNA 분자의 영역을 포함한다. 기타 경우에, DNA-표적화 RNA의 단백질-결합 세그먼트(이하 기술됨)는 상보성 영역에 따라 하이브리드화되는 2개의 별도의 분자를 포함한다. 예시적인 비제한적인 예로서, 두 개의 별도의 분자를 포함하는 DNA-표적화 RNA의 단백질-결합 세그먼트는 (i) 길이 100 개의 염기쌍인 제 1 RNA 분자의 염기쌍 40 내지 75 및 (ii) 길이 50 개의 염기쌍인 제 2 RNA 분자의 염기쌍 10 내지 25를 포함한다. "세그먼트"의 정의는, 특별한 문맥에서 달리 구체적으로 정의되지 않는 한, 총 염기쌍의 특정 수에 국한되지 않고, 주어진 RNA 분자로부터 염기쌍의 임의의 특정 수에 국한되지 않고, 복합체 내의 별도의 분자의 특정 수에 국한되지 않고, 임의의 총 길이인 RNA 분자의 영역을 포함할 수 있고, 기타 분자에 대한 상보성을 갖는 영역을 포함할 수 있거나 포함하지 않을 수 있다.
DNA-표적화 세그먼트(또는 "DNA-표적화 서열")은 표적 DNA 내의 특이적 서열에 상보적인 뉴클레오타이드 서열(표적 DNA의 상보적 가닥)을 포함한다. 단백질 결합 세그먼트(또는 "단백질 결합 서열")은 부위 지향적 변형 폴리펩타이드와 상호작용한다. 부위 지향적 변형 폴리펩타이드가 Cas9 또는 Cas9 관련 폴리펩타이드(이하 보다 상세히 기재됨)일 경우, 표적 DNA의 부위 지향적 절단은 (i) DNA-표적화 RNA 및 표적 DNA 사이의 염기 짝짓기 상보성 및 (ii) 표적 DNA 중의 짧은 모티프(프로토스페이서 인접 모티프(PAM)로서 칭명됨) 둘 다에 의해 결정된 위치에서 일어난다.
당해 DNA-표적화 RNA의 단백질 결합 세그먼트는 서로 하이브리드화하여 이중 가닥 RNA 듀플렉스(dsRNA 듀플렉스)를 형성하는 뉴클레오타이드의 두 개의 상보성 스트레치를 포함한다.
일부 구현예에서, 당해 핵산(예: DNA-표적화 RNA, DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산; 부위 지향적 폴리펩타이드를 인코딩하는 핵산 등)은 추가의 바람직한 특성(예: 변형되거나 조절된 안정성, 준세포 표적화; 추적, 예를 들면, 형광 라벨; 단백질 또는 단백질 복합체용 결합 부위 등)을 제공하는 변형 또는 서열을 포함한다. 비제한적인 예들은 5' 캡(예: 7-메틸구아닐레이트 캡(m7G)); 3' 폴리아데닐화 꼬리(즉, 3' 폴리(A) 꼬리); 리보스위치 서열(예: 단백질 및/또는 단백질 복합체에 의한 조절된 안정성 및/또는 조절된 접근성을 허용하는); 안정성 제어 서열; dsRNA 듀플렉스(즉, 헤어핀)을 형성하는 서열; RNA를 준세포 위치(예: 핵, 미토콘드리아, 엽록체 등)에서 표적화하는 변형 또는 서열; 추적(예: 형광성 분자에 직접 접합, 형광 검출을 촉진시키는 잔기에 접합, 형광 검출을 허용하는 서열 등)을 제공하는 변형 또는 서열; 단백질(예: 전사 활성제, 전사 억제인자, DNA 메틸트랜스퍼라제, DNA 데메틸라제, 히스톤 아세틸트랜스퍼라제, 히스톤 데아세틸라제 등을 포함하는 DNA에 작용하는 단백질)에 결합 부위를 제공하는 변형 또는 서열; 및 이들의 조합을 포함한다.
일부 구현예에서, DNA-표적화 RNA는 상기 기재된 임의의 특징을 제공하는 5' 또는 3' 말단에 추가의 세그멘트를 포함한다. 예를 들면, 적합한 제 3 세그먼트는 5' 캡(예: 7-메틸구아닐레이트 캡(m7G)); 3 폴리아데닐화 꼬리(즉, 3' 폴리(A) 꼬리); 리보스위치 서열(예: 단백질 및/또는 단백질 복합체에 의한 조절된 안정성 및/또는 조절된 접근성을 허용하는); 안정성 제어 서열; dsRNA 듀플렉스(즉, 헤어핀)를 형성하는 서열; RNA를 준세포 위치(예: 핵, 미토콘드리아, 엽록체 등)에서 표적화하는 서열; 추적(예: 형광성 분자에 직접 접합, 형광 검출을 촉진시키는 잔기에 접합, 형광 검출을 허용하는 서열 등)을 제공하는 변형 또는 서열; 단백질(예: 전사 활성제, 전사 억제인자, DNA 메틸트랜스퍼라제, DNA 데메틸라제, 히스톤 아세틸트랜스퍼라제, 히스톤 데아세틸라제 등을 포함하는 DNA에 작용하는 단백질)에 결합 부위를 제공하는 변형 또는 서열; 및 이들의 조합을 포함할 수 있다.
당해 DNA-표적화 RNA 및 당해 부위 지향적 변형 폴리펩타이드(즉, 부위 지향적 폴리펩타이드)는 복합체를 형성한다(즉, 비공유 상호작용을 통해 결합한다). DNA-표적화 RNA는 표적 DNA의 서열에 상보적인 뉴클레오타이드 서열을 포함함으로써 복합체에 표적 특이성을 제공한다. 복합체의 부위 지향적 변형 폴리펩타이드는 부위-특이적 활성을 제공한다. 환언하면, 부위 지향적 변형 폴리펩타이드는 DNA-표적화 RNA의 단백질-결합 세그먼트과의 회합에 의해 표적 DNA 서열(예: 염색체 핵산 중의 표적 서열; 염색체외 핵산, 예를 들면, 에피솜 핵산, 미니써클 등 중의 표적 서열; 미토콘드리아 핵산 중의 표적 서열; 엽록체 핵산 중의 표적 서열; 플라스미드 중의 표적 서열 등)에 유도된다.
일부 구현예에서, 당해 DNA-표적화 RNA는 두 개의 별도의 RNA 분자(RNA 폴리뉴클레오타이드: "활성제-RNA" 및 "타겟터-RNA", 이하 참조)를 포함하고, 본원에서 "이중-분자 DNA-표적화 RNA" 또는 "두-분자 DNA-표적화 RNA"로서 칭명된다. 기타 구현예에서, 당해 DNA-표적화 RNA는 단일 RNA 분자(단일 RNA 폴리뉴클레오타이드)이고, 본원에서 "단일-분자 DNA-표적화 RNA", "단일-가이드 RNA" 또는 "sgRNA"로서 칭명된다. 용어 "DNA-표적화 RNA" 또는 "gRNA"는 포괄적이어서, 이중-분자 DNA-표적화 RNA 및 단일-분자 DNA-표적화 RNA(즉, sgRNA) 둘 다를 언급한다.
예시적인 두-분자 DNA-표적화 RNA는 crRNA-형("CRISPR RNA" 또는 "타겟터-RNA" 또는 "crRNA" 또는 "crRNA 반복체") 분자 및 상응하는 tracrRNA-형("트랜스-작용성 CRISPR RNA" 또는 "활성제-RNA" 또는 "tracrRNA") 분자를 포함한다. crRNA-형 분자(타겟터-RNA)는 DNA-표적화 RNA의 DNA-표적화 세그먼트(단일 가닥) 및 DNA-표적화 RNA의 단백질-결합 세그먼트의 dsRNA 듀플렉스의 1/2를 형성하는 뉴클레오타이드의 스트레치("듀플렉스 형성 세그먼트") 모두를 포함한다. 상응하는 tracrRNA-형 분자(활성제-RNA)는 DNA-표적화 RNA의 단백질-결합 세그먼트의 dsRNA 듀플렉스의 나머지 1/2를 형성하는 뉴클레오타이드의 스트레치(듀플렉스 형성 세그먼트)를 포함한다. 환언하면, crRNA-형 분자의 뉴클레오타이드의 스트레치는 tracrRNA-형 분자의 뉴클레오타이드의 스트레치에 상보적이고, 이와 하이브리드화하여 DNA-표적화 RNA의 단백질-결합 도메인의 dsRNA 듀플렉스를 형성한다. 이와 같이, 각각의 crRNA-형 분자는 상응하는 tracrRNA-형 분자를 갖는다고 할 수 있다. crRNA-형 분자는 추가로 단일 가닥 DNA-표적화 세그먼트를 제공한다. 따라서, crRNA-형 분자 및 tracrRNA-형 분자(상응하는 쌍으로서)가 하이브리드화하여 DNA-표적화 RNA를 형성한다. 주어진 crRNA 또는 tracrRNA 분자의 정확한 서열은 RNA 분자가 발견되는 종의 특징이다. 다양한 crRNA 및 tracrRNA는 도 8에서 상응하는 상보적 쌍으로 묘사된다. 당해 이중-분자 DNA-표적화 RNA는 임의의 상응하는 crRNA 및 tracrRNA 쌍을 포함할 수 있다. 당해 이중-분자 DNA-표적화 RNA는 임의의 상응하는 crRNA 및 tracrRNA 쌍을 포함할 수 있다.
용어 "활성제-RNA"는 이중-분자 DNA-표적화 RNA의 tracrRNA-형 분자를 의미하기 위해 본원에서 사용된다. 용어 "타겟터-RNA"는 이중-분자 DNA-표적화 RNA의 crRNA-형 분자를 의미하기 위해 본원에서 사용된다. 용어 "듀플렉스-형성 세그먼트"은 상응하는 활성제-RNA 또는 타겟터-RNA 분자의 뉴클레오타이드의 스트레치로 하이브리드화함으로써 dsRNA 듀플렉스의 형성에 기여하는 활성제-RNA 또는 타겟터-RNA의 뉴클레오타이드의 스트레치를 의미하기 위해 본원에서 사용된다. 환언하면, 활성제-RNA는 상응하는 타겟터-RNA의 듀플렉스-형성 세그먼트에 상보적인 듀플렉스-형성 세그먼트를 포함한다. 이와 같이, 활성제-RNA는 듀플렉스-형성 세그먼트를 포함하는 반면, 타겟터-RNA는 듀플렉스-형성 세그먼트 및 DNA-표적화 RNA의 DNA-표적화 세그먼트 둘 다를 포함한다. 따라서, 당해 이중-분자 DNA-표적화 RNA는 임의의 상응하는 활성제-RNA 및 타겟터-RNA 쌍으로 이루어질 수 있다.
본원에 사용된 "숙주 세포"는 생체내 또는 시험관내 진핵 세포, 원핵 세포(예: 박테리아 또는 고세균 세포), 또는 단세포 독립체로서 배양된 다중세포 유기체(예: 세포주)로부터의 세포를 나타내고, 이 진핵 또는 원핵 세포가 핵산을 위한 수령체로서 사용될 수 있거나 사용되었고, 핵산에 의해 형질전환된 본래 세포의 자손을 포함한다. 단일 세포의 자손은 천연, 우발적 또는 계획적 돌연변이에 기인하여, 원래 부모와 형태 또는 게놈 또는 총 DNA 보체가 반드시 완전히 동일할 필요가 없을 수 있는 것으로 이해된다. "재조합 숙주 세포"(또한 "유전적으로 변형된 숙주 세포"로서 칭명됨)는 이종기원 핵산, 예를 들면, 발현 벡터가 도입된 숙주 세포이다. 예를 들면, 당해 박테리아 숙주 세포는 외인성 핵산(예: 플라스미드 또는 재조합 발현 벡터)의 적당한 박테리아 숙주 세포에 도입함으로써 유전적으로 변형된 박테리아 숙주 세포이고, 당해 진핵 숙주 세포는 외인성 핵산의 적당한 진핵 숙주 세포에 도입시킴으로써 유전적으로 변형된 진핵 숙주 세포(예: 포유동물 생식 세포)이다.
용어 "줄기 세포"는 자기 갱신하고 분화된 세포형을 생성하는 능력 모두를 갖는 세포(예: 식물 줄기 세포, 척추동물 줄기 세포)를 의미하기 위해 본원에서 사용된다(참조: Morrison 등 (1997) Cell 88:287-298). 세포 개체발생의 맥락에서, 형용사 "분화된" 또는 "분화하는"은 상대적인 용어이다. "분화된 세포"는 비교되는 세포보다 발달 경로 아래로 추가로 진행되는 세포이다. 따라서, 만능 줄기 세포(이하 기재됨)는 계통-제한된 전구 세포(예: 중배엽 줄기 세포)로 분화시킬 수 있고, 이는 결국 제한된 세포(예: 뉴런 조상세포)로 분화시킬 수 있고, 이는 말기 세포(즉, 말단 분화된 세포, 예를 들면, 뉴런, 심근세포 등)로 분화시킬 수 있고, 이는 특정 조직 형태에서 독특한 역할을 하고, 추가로 증식시키는 용량을 유지시킬 수 있거나 유지시키지 않을 수 있다. 줄기 세포는 특이적 마커(예: 단백질, RNA 등)의 존재 및 특이적 마커의 부재 둘 다에 의해 특성화될 수 있다. 줄기 세포는 또한 시험관내 및 생체내 둘 다에서 기능적 검정, 특히 다중 분화된 자손을 발생시키는 줄기 세포의 능력에 관련된 검정에 의해 확인될 수 있다.
목적하는 줄기 세포는 만능 줄기 세포(PSC)를 포함한다. 용어 "만능 줄기 세포" 또는 "PSC"는 유기체의 모든 세포형을 생성할 수 있는 줄기 세포를 의미하기 위해 본원에서 사용된다. 따라서, PSC는 유기체의 모든 배엽층(예: 척추동물의 내배엽, 중배엽 및 외배엽)의 세포를 발생시킬 수 있다. 만능 세포는 기형종을 형성할 수 있고, 살아있는 유기체의 외배엽, 중배엽 또는 내배엽 조직에 기여할 수 있다. 식물의 만능 줄기 세포는 식물의 모든 세포형(예: 뿌리, 줄기, 잎 등의 세포)을 발생시킬 수 있다.
동물의 PSC는 다수의 상이한 방식으로 유도될 수 있다. 예를 들면, 배아 줄기 세포(ESC)는 배아의 내부 세포 덩어리부터 유도되고(참조: Thomson 등 Science. 1998 Nov 6;282(5391):1145-7), 반면에 유도된 만능 줄기 세포(iPCS)는 체세포로부터 유도된다(참조: Takahashi 등 Cell. 2007 Nov 30;131(5):861-72; Takahashi 등 Nat Protoc. 2007;2(12):3081-9; Yu 등 Science. 2007 Dec 21;318(5858):1917-20. Epub 2007 Nov 20). 용어 PSC가 그의 유도와 무관하게 만능 줄기 세포를 의미하기 때문에, 용어 PSC는 용어 ESC 및 iPCS 뿐만 아니라 PSC의 또 하나의 예인 용어 배아 생식 줄기 세포(EGSC)를 포함한다. PSC는 확립된 세포주의 형태일 수 있고, 그들은 1차 배아 조직으로부터 직접 수득될 수 있거나, 그들은 체세포로부터 유도될 수 있다. PSC는 본원에서 기재된 방법의 표적 세포일 수 있다.
"배아 줄기 세포"(ESC)란, 배아로부터, 전형적으로 배반포의 내부 세포 덩어리로부터 단리된 PSC를 의미한다. ESC 라인, 예를 들면, hESBGN-01, hESBGN-02, hESBGN-03, hESBGN-04 (BresaGen, Inc.); HES-1, HES-2, HES-3, HES-4, HES-5, HES-6 (ES Cell International); Miz-hES1 (MizMedi Hospital-Seoul National University); HSF-1, HSF-6 (University of California at San Francisco); 및 H1, H7, H9, H13, H14 (Wisconsin Alumni Research Foundation (WiCell Research Institute))가 NIH 인간 배아 줄기 세포 등록에 열거된다. 목적하는 줄기 세포는 또한 다른 영장류로부터의 배아 줄기 세포, 예를 들면, 붉은 털 원숭이 줄기 세포 및 마모셋 줄기 세포를 포함한다. 줄기 세포는 임의의 포유동물 종, 예를 들면, 인간, 말, 소, 돼지, 갯과, 고양이, 설치류, 예를 들면, 마우스, 래트, 햄스터, 영장류 등으로부터 수득될 수 있다(참조: Thomson 등 (1998) Science 282:1145; Thomson 등 (1995) Proc. Natl. Acad. Sci USA 92:7844; Thomson 등 (1996) Biol. Reprod. 55:254; Shamblott 등, Proc. Natl. Acad. Sci. USA 95:13726, 1998). 배양물에서, ESC는 전형적으로 거대한 핵-세포질 비, 규정된 경계 및 두드러진 핵인을 갖는 평평한 콜로니로서 성장한다. 또한, ESC는 SSEA-1이 아니라 SSEA-3, SSEA-4, TRA-1-60, TRA-1-81 및 알칼리성 포스파타제를 발현시킨다. ESC를 생성하고 특성화하는 방법의 예는, 예를 들면, 명세서가 본원에 참조로 인용된 미국 특허 제7,029,913호, 미국 특허 제5,843,780호 및 미국 특허 제6,200,806호에서 찾을 수 있다. 미분화된 형태로 hESC를 증식시키는 방법은 WO 99/20741, WO 01/51616 및 WO 03/020920에 기재되어 있다.
"배아 생식 줄기 세포"(EGSC) 또는 "배아 생식 세포" 또는 "EG 세포"는 생식 세포 및/또는 생식 세포 조상세포, 예를 들면, 원시 생식 세포로부터 유도된 PSC, 즉 정자와 알이 되는 것들을 의미한다. 배아 생식 세포(EG 세포)는 상기에서 기재된 바와 같은 배아 줄기 세포와 유사한 특성을 갖는 것으로 생각된다. EG 세포를 생성하고 특성화하는 방법의 예는, 예를 들면, 명세서가 본원에 참조로 인용된 문헌(참조: 미국 특허 제7,153,684호, Matsui, Y., 등, (1992) Cell 70:841; Shamblott, M., 등 (2001) Proc. Natl. Acad. Sci. USA 98: 113; Shamblott, M., 등 (1998) Proc. Natl. Acad. Sci. USA, 95:13726; 및 Koshimizu, U., 등 (1996) Development, 122:1235)에서 찾을 수 있다.
"유도된 만능 줄기 세포" 또는 "iPSC"란, PSC가 아닌 세포로부터(즉, 이것이 PSC에 상대적으로 분화된 세포로부터) 유도된 PSC를 의미한다. iPSC는 말단 분화된 세포를 포함하는 다중 상이한 세포형으로부터 유도될 수 있다. iPSC는 거대한 핵-세포질 비, 규정된 경계 및 두드러진 핵을 갖는 평평한 콜로니로서 성장하는 ES 세포-형 형태를 가지고 있다. 또한, iPSC는 알칼리성 포스파타제, SSEA3, SSEA4, Sox2, Oct3/4, Nanog, TRA160, TRA181, TDGF 1, Dnmt3b, FoxD3, GDF3, Cyp26a1, TERT 및 zfp42를 포함하지만, 이에 국한되지 않는 당해 기술 분야의 숙련가에게 공지된 하나 이상의 중요 전문화능 마커를 발현시킨다. iPSC를 생성하고 특성화하는 방법의 예는, 예를 들면, 명세서가 본원에 참조로 인용된 미국 특허 공보 제US20090047263호, 제US20090068742호, 제US20090191159호, 제US20090227032호, 제US20090246875호 및 제US20090304646호에서 찾을 수 있다. 일반적으로, iPSC를 생성하기 위해, 체세포에 체세포를 만능 줄기 세포가 되도록 재프로그로밍하는 것으로 당해 기술 분야에 공지된 재프로그래밍 인자(예: Oct4, SOX2, KLF4, MYC, Nanog, Lin28 등)가 제공된다.
"체세포"란 실험 조작의 부재하에 유기체에서 대개 모든 유형의 세포를 발생시키지 않는 유기체 중의 임의의 세포를 의미한다. 환언하면, 체세포는, 이들이 신체의 모든 3개의 배엽층, 즉, 외배엽, 중배엽 및 내배엽의 세포를 자연적으로 생성하지 않도록 충분히 분화된 세포이다. 예를 들면, 체세포는 뉴런 및 신경 조상세포 둘 다를 포함할 수 있고, 이중 후자는 중추신경계의 모든 또는 일부 세포형을 자연적으로 발생시킬 수 있지만, 중배엽 또는 내배엽 계통의 세포를 발생시킬 수 없다.
"유사분열 세포"란 유사분열을 경험하는 세포를 의미한다. 유사분열은 진핵 세포가 이의 핵 중의 염색체를 2개의 별도의 핵 중의 2개의 동일한 세트로 분리하는 과정이다. 이는 일반적으로 세포질분열 직후에 따르고, 이는 핵, 세포질, 소 기관 및 세포막을 이러한 세포성 성분의 거의 동일한 부분을 함유하는 두 세포로 분할한다.
"후-유사분열 세포"란, 유사분열로부터 방출된 세포를 의미하고, 즉, 이는 "정지되어" 있고, 즉 이는 더 이상 분열을 경험하지 않는다. 이 정지 상태는 일시적, 즉 가역적일 수 있거나, 이는 영구적일 수 있다.
"감수분열 세포"란 감수분열을 경험하는 세포를 의미한다. 감수분열은 세포가 생식체 또는 포자를 생성하기 위해 이의 핵 물질을 분할하는 과정이다. 유사분열과 다르게, 감수분열에서 염색체는 염색체 사이의 유전적 물질을 혼합하는 재조합 단계를 경험한다. 추가로, 감수분열의 결과는 유사분열로부터 생성된 2개의 (유전적으로 동일한) 2배체 세포와 비교하여, 4개의 (유전적으로 독특한) 반수체 세포이다.
"제조합"이란, 두 폴리뉴클레오타이드 사이의 유전적 정보의 교환 과정을 의미한다. 본원에 사용된 "상동성-유도된 치유(HDR)"는, 예를 들면, 세포 중에서 이중-가닥 파괴의 치유 동안 발생하는 특화된 형태 DNA 치유를 의미한다. 이 과정은 뉴클레오타이드 서열 상동성을 필요로 하고, "표적" 분자(즉, 이중-가닥 파괴를 경험한 것)의 템플레이트 치유하기 위해 "공여체" 분자를 사용하고, 공여체로부터 표적으로 유전적 정보의 전달을 유도한다. 상동성-유도된 치유는, 공여체 폴리뉴클레오타이드가 표적 분자와 상이하고, 공여체 폴리뉴클레오타이드의 서열의 일부 또는 모두가 표적 DNA에 편입되는 경우, 표적 분자 서열의 변경(예: 삽입, 결실, 돌연변이)을 유도할 수 있다. 일부 구현예에서, 공여체 폴리뉴클레오타이드, 공여체 폴리뉴클레오타이드의 일부, 공여체 폴리뉴클레오타이드의 카피 또는 공여체 폴리뉴클레오타이드의 카피의 일부는 표적 DNA에서 통합한다.
"비-상동 말단 결합(NHEJ)"이란, (치유를 유도하기 위해 상동 서열을 필요로 하는 상동성-유도된 치유와 대조적으로) 상동 템플레이트에 대한 요구 없이 파괴 말단 서로의 직접 결찰로 DNA 중의 이중-가닥 파괴의 치유를 의미한다. NHEJ는 흔히 이중-가닥 파괴 부위 근처의 뉴클레오타이드 서열의 손실(결실)을 유도한다.
용어 "치료", "치료하는" 등은 일반적으로 목적하는 약리학적 및/또는 생리적 효과를 수득함을 의미하기 위해 본원에서 사용된다. 상기 효과는 질환 또는 이의 증상을 완전히 또는 부분적으로 예방하는 관점에서 예방적일 수 있고/있거나 질환 및/또는 이 질환에 기인하는 역효과를 부분적 또는 완전한 치유의 관점에서 치료적일 수 있다. 본원에 사용된 "치료"는 포유동물에서 질환 또는 증상의 임의 치료를 포함하고, (a) 아직 질환 또는 증상을 갖는 것으로 진단되지 않았지만 그것에 걸리기 쉬울 수 있는 대상체에서 질환 또는 증상이 발생하는 것을 방지하고; (b) 질환 또는 증상을 억제하고, 즉 이의 발생을 저지하거나; 또는 (c) 질환을 완화시키고, 즉, 질환의 퇴행을 일으킴을 포함한다. 치료제는 질환 또는 손상의 발병 전, 동안 또는 후에 투여될 수 있다. 치료가 환자의 바람직하지 않은 임상적 증상을 안정화시키거나 감소시키는 진행 중인 질환의 치료가 특히 중요하다. 이러한 치료는 바람직하게는 침범된 조직에서 기능의 완전한 손실 전에 수행된다. 당해 요법은 바람직하게는 질환의 증상 단계 동안, 일부 경우에는 질환의 증상 단계 후에 투여된다.
용어 "개체", "대상체", "숙주" 및 "환자"는 본원에서 상호교환적으로 사용되고, 진단, 치료 및 요법을 목적으로 하는 임의의 포유동물 대상체, 특히 인간을 의미한다.
분자 및 세포 생화학에서 일반적인 방법은 명세서가 본원에 참조로 인용된 다음과 같은 표준 교과서에서 찾을 수 있다: Molecular Cloning: A Laboratory Manual, 3rd Ed. (Sambrook 등, HaRBor Laboratory Press 2001); Short Protocols in Molecular Biology, 4th Ed. (Ausubel 등 eds., John Wiley & Sons 1999); Protein Methods (Bollag 등, John Wiley & Sons 1996); Nonviral Vectors for Gene Therapy (Wagner 등 eds., Academic Press 1999); Viral Vectors (Kaplift & Loewy eds., Academic Press 1995); Immunology Methods Manual (I. Lefkovits ed., Academic Press 1997); 및 Cell and Tissue Culture: Laboratory Procedures in Biotechnology (Doyle & Griffiths, John Wiley & Sons 1998).
본 발명이 추가로 기재되기 전에, 본 발명은 물론 변할 수 있기 때문에, 기재된 특정 구현예에 국한되지 않는 것으로 이해되어야 한다. 또한, 본원에 사용된 용어는 단지 특정 구현예를 기술하기 위한 것이고, 본 발명의 범위가 단지 첨부된 특허청구범위에 의해서만 제한되기 때문에, 제한하고자 하지 않는다고 이해되어야 한다.
값의 범위가 제공될 경우, 각각의 개재 값, 문맥이 명백하게 달리 지시하지 않는 한 그 범위의 하한치의 단위의 1/10까지, 그 범위의 상한치와 하한치 사이 및 그 기술된 범위 내의 임의의 다른 언급된 또는 개입 값이 본 발명 내에 포함된다고 이해된다. 이들 더 작은 범위의 상한치 및 하한치는 독립적으로 더 작은 범위에 포함될 수 있고, 또한 언급된 범위에서 임의의 특이적으로 제외된 한계까지 본 발명 대상체내에 포함된다. 언급된 범위가 한계치 중의 하나 또는 둘 다를 포함할 경우, 포함된 한계치 중의 하나 또는 둘 다를 제외하는 범위도 또한 본 발명에 포함된다.
특정 범위는 본원에서 용어 "약"이 선행되는 수치 값과 함께 제공된다. 용어 "약"은 그것이 선행하는 정확한 수 뿐만 아니라 그 용어가 선행하는 수에 근접하거나 대략적인 수에 대한 문자그대로의 지지를 제공하기 위해 본원에서 사용된다. 수가 특이적으로 인용된 수에 근접하거나 대략적인지를 결정하는데 있어서, 근접하거나 또는 대략적인 인용되지 않은 수는 그것이 제공되는 문맥에 있어서, 특이적으로 인용된 수의 실질적인 등가물을 제공하는 수일 수 있다.
달리 정의되지 않으면, 본원에 사용된 모든 기술 및 과학 용어들은 본 발명이 속하는 당해 분야의 숙련가에 의해 전형적으로 이해되는 바와 동일한 의미를 갖는다. 본원에서 기술된 것들과 유사하거나 등가인 임의의 방법 및 물질이 또한 본 발명의 실시 또는 시험에 사용될 수 있지만, 바람직한 방법 및 물질은 이하 기술된다. 본원에서 언급된 모든 공보들은 인용된 공보와 관련하여 방법 및/또는 물질을 나타내고 기술하기 위해 본원에 참조로 인용된다.
본 명세서에 인용된 모든 공보 및 특허는 각각 개별적 공보 또는 특허가 본원에 참조로 인용된 것으로 구체적으로 및 개별적으로 나타낸 것처럼 본원에 참조로 인용되고, 인용된 공보와 관련하여 방법 및/또는 물질을 나타내고 기술하기 위해 본원에 참조로 인용된다. 임의의 공보의 인용은 출원일 이전의 이의 개시 내용을 위해서이고, 본 발명이 선행 발명에 의해 이러한 공보에 선행할 자격이 없음을 승인하는 것으로서 해석되어서는 안된다. 추가로, 제공된 공고일은 독립적으로 확인될 필요가 있을 수 있는 실제 공고일과 상이할 수 있다.
본원 및 첨부된 특허청구범위에서 사용된 바와 같이, 단수 형태 "a", "an" 및 "the"는 문맥이 명확히 달리 지시하지 않는 한 복수 지시대상을 포함한다는 것이 주시된다. 따라서, 예를 들면, "폴리뉴클레오타이드"에 대한 참조는 복수의 이러한 폴리뉴클레오타이드를 포함하고, "폴리펩타이드"에 대한 참조는 하나 이상의 폴리펩타이드 및 당해 기술 분야의 숙련가에게 공지된 이의 등가물 등에 대한 참조를 포함한다. 또한, 특허청구범위가 임의의 선택적 요소를 제외하도록 드래프트될 수 있다는 것이 주시된다. 이와 같이, 이 서술은 청구항 요소의 설명 또는 "음성" 한계의 사용과 관련하여 "유일하게", "단지" 등과 같은 이러한 배타적인 용어의 사용에 대한 선행된 기준으로서 작용하도록 의도된다.
명쾌함을 위해, 개별적 구현예의 문맥에서 기재된 본 발명의 특정의 특징은 또한 단일 구현예에 조합으로 제공될 수 있다고 인정된다. 반대로, 간결함을 위해 단일 구현예의 문맥에서 기재된 본 발명의 다양한 특징은 또한 별도로 또는 임의의 적합한 아-조합으로 제공될 수 있다. 본 발명에 속하는 구현예의 모든 조합은 구체적으로 본 발명에 의해 포함되고, 마치 각각 및 모든 조합이 개별적으로 명백하게 개시된 것처럼 본원에서 개시된다. 또한, 다양한 구현예의 모든 아-조합 및 이의 요소는 또한 본 발명에 의해 구체적으로 포함되고, 마치 각각 및 모든 이러한 아-조합이 개별적으로 명백하게 본원에서 개시된 것처럼 본원에서 개시된다.
본원에서 논의된 공보들은 단지 본원의 출원일 이전의 이들의 개시내용을 위해 제공된다. 본원에서 어떤 것도 본 발명이 선행 발명에 의해 이러한 공보를 선행할 자격이 없음을 승인하는 것으로서 해석되어서는 안된다. 게다가, 제공된 공고일은 독립적으로 확인될 필요가 있을 수 있는 실제 공고일과 상이할 수 있다.
상세한 설명 - 파트 I
당해 개시내용은 표적 서열을 포함하는 DNA-표적화 RNA를 제공하고, 변형 폴리펩타이드와 함께, 표적 DNA 및/또는 표적 DNA와 연관된 폴리펩타이드의 부위 특이적인 변형을 제공한다. 당해 개시내용은 추가로, 부위 특이적인 변형 폴리펩타이드를 제공한다. 당해 개시내용은 추가로, 표적 DNA 및/또는 표적 DNA와 연관된 폴리펩타이드의 부위 특이적인 변형 방법을 제공한다. 당해 개시내용은 표적 세포에서 표적 핵산의 전사를 조절하는 방법을 제공하고, 일반적으로 상기 표적 핵산을 효소적으로 불활성 Cas9 폴리펩타이드 및 DNA-표적화 RNA와 접촉시키는 것을 수반한다. 당해 방법을 수행하기 위한 키트 및 조성물이 또한 제공된다. 당해 개시내용은 Cas9를 생산하는 유전적으로 변형된 세포; 및 Cas9 이식유전자 비-인간 다중세포 유기체를 제공한다.
핵산
DNA - 표적화 RNA
당해 개시내용은 표적 DNA 내에서 특이적 표적 서열에 대해에 대해 연관된 폴리펩타이드 (예를 들면, 부위 지향적 변형 폴리펩타이드)의 활성을 겨냥하는 DNA-표적화 RNA를 제공한다. 당해 DNA-표적화 RNA는 하기를 포함한다: 제 1 분절 ("DNA-표적화 분절" 또는 "DNA-표적화 서열"로도 본원에서 칭함) 및 제 2 분절 ("단백질-결합 세그먼트" 또는 "단백질-결합 서열"로도 본원에서 칭함).
DNA - 표적화 RNA 의 DNA - 표적화 분절
당해 DNA-표적화 RNA의 DNA-표적화 분절은 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함한다. 환언하면, 당해 DNA-표적화 RNA의 DNA-표적화 분절은 하이브리드화 (즉, 염기 짝짓기)를 통해 서열-특이적 방식으로 표적 DNA와 상호작용한다. 이와 같이, DNA-표적화 분절의 뉴클레오타이드 서열은 DNA-표적화 RNA 및 표적 DNA가 상호작용할 표적 DNA 내에서 변하고 위치를 결정할 수 있다. 당해 DNA-표적화 RNA의 DNA-표적화 분절은 (예를 들면, 유전공학에 의해) 변형되어 표적 DNA 내에서 임의의 원하는 서열을 혼성화할 수 있다.
DNA-표적화 분절은 그 길이가 약 12 뉴클레오타이드 내지 약 100 개의 뉴클레오타이드일 수 있다. 예를 들면, DNA-표적화 분절은 그 길이가 약 12 뉴클레오타이드 (nt) 내지 약 80 nt, 약 12 nt 내지 약 50nt, 약 12 nt 내지 약 40 nt, 약 12 nt 내지 약 30 nt, 약 12 nt 내지 약 25 nt, 약 12 nt 내지 약 20 nt, 또는 약 12 nt 내지 약 19 nt일 수 있다. 예를 들면, DNA-표적화 분절은 그 길이가 약 19 nt 내지 약 20 nt, 약 19 nt 내지 약 25 nt, 약 19 nt 내지 약 30 nt, 약 19 nt 내지 약 35 nt, 약 19 nt 내지 약 40 nt, 약 19 nt 내지 약 45 nt, 약 19 nt 내지 약 50 nt, 약 19 nt 내지 약 60 nt, 약 19 nt 내지 약 70 nt, 약 19 nt 내지 약 80 nt, 약 19 nt 내지 약 90 nt, 약 19 nt 내지 약 100 nt, 약 20 nt 내지 약 25 nt, 약 20 nt 내지 약 30 nt, 약 20 nt 내지 약 35 nt, 약 20 nt 내지 약 40 nt, 약 20 nt 내지 약 45 nt, 약 20 nt 내지 약 50 nt, 약 20 nt 내지 약 60 nt, 약 20 nt 내지 약 70 nt, 약 20 nt 내지 약 80 nt, 약 20 nt 내지 약 90 nt, 또는 약 20 nt 내지 약 100 nt일 수 있다. 표적 DNA의 뉴클레오타이드 서열 (표적 서열)에 대해 상보적인 DNA-표적화 분절의 뉴클레오타이드 서열 (DNA-표적화 서열)은 그 길이가 적어도 약 12 nt일 수 있다. 예를 들면, 표적 DNA의 표적 서열에 대해 상보적인 DNA-표적화 분절의 DNA-표적화 서열은 그 길이가 적어도 약 12 nt, 적어도 약 15 nt, 적어도 약 18 nt, 적어도 약 19 nt, 적어도 약 20 nt, 적어도 약 25 nt, 적어도 약 30 nt, 적어도 약 35 nt 또는 적어도 약 40 nt일 수 있다. 예를 들면, 표적 DNA의 표적 서열에 대해 상보적인 DNA-표적화 분절의 DNA-표적화 서열은 그 길이가 약 12 뉴클레오타이드 (nt) 내지 약 80 nt, 약 12 nt 내지 약 50nt, 약 12 nt 내지 약 45 nt, 약 12 nt 내지 약 40 nt, 약 12 nt 내지 약 35 nt, 약 12 nt 내지 약 30 nt, 약 12 nt 내지 약 25 nt, 약 12 nt 내지 약 20 nt, 약 12 nt 내지 약 19 nt, 약 19 nt 내지 약 20 nt, 약 19 nt 내지 약 25 nt, 약 19 nt 내지 약 30 nt, 약 19 nt 내지 약 35 nt, 약 19 nt 내지 약 40 nt, 약 19 nt 내지 약 45 nt, 약 19 nt 내지 약 50 nt, 약 19 nt 내지 약 60 nt, 약 20 nt 내지 약 25 nt, 약 20 nt 내지 약 30 nt, 약 20 nt 내지 약 35 nt, 약 20 nt 내지 약 40 nt, 약 20 nt 내지 약 45 nt, 약 20 nt 내지 약 50 nt, 또는 약 20 nt 내지 약 60 nt일 수 있다. 표적 DNA의 뉴클레오타이드 서열 (표적 서열)에 대해 상보적인 DNA-표적화 분절의 뉴클레오타이드 서열 (DNA-표적화 서열)은 그 길이가 적어도 약 12 nt일 수 있다.
일부 경우에서, 표적 DNA의 표적 서열에 대해 상보적인 DNA-표적화 분절의 DNA-표적화 서열은 그 길이가 20 개의 뉴클레오타이드이다. 일부 경우에서, 표적 DNA의 표적 서열에 대해 상보적인 DNA-표적화 분절의 DNA-표적화 서열은 그 길이가 19 개의 뉴클레오타이드이다.
The DNA-표적화 분절의 DNA-표적화 서열과 표적 DNA의 표적 서열 사이의 상보성 %는 적어도 60% (예를 들면, 적어도 65%, 적어도 70%, 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 적어도 97%, 적어도 98%, 적어도 99%, 또는 100%)일 수 있다. 일부 경우에서, DNA-표적화 분절의 DNA-표적화 서열과 표적 DNA의 표적 서열 사이의 상보성 %은 표적 DNA의 상보적 가닥의 표적 서열 7 개의 인접 5'-최대 뉴클레오타이드에 대해 100%이다. 일부 경우에서, DNA-표적화 분절의 DNA-표적화 서열과 표적 DNA의 표적 서열 사이의 상보성 %은 약 20 개의 인접 뉴클레오타이드에 대해 적어도 60%이다. 일부 경우에서, DNA-표적화 분절의 DNA-표적화 서열과 표적 DNA의 표적 서열 사이의 상보성 %은 표적 DNA의 상보적 가닥의 표적 서열의 14 개의 인접 5'-최대 뉴클레오타이드에 대해 100%이고 나머지에 대해서는 0% 정도로 낮다. 그와 같은 경우에, DNA-표적화 서열은 그 길이가 14 개의 뉴클레오타이드인 것으로 간주될 수 있다 (참고 도 12d-e). 일부 경우에서, DNA-표적화 분절의 DNA-표적화 서열과 표적 DNA의 표적 서열 사이의 상보성 %은 표적 DNA의 상보적 가닥의 표적 서열의 7 개의 인접 5'-최대 뉴클레오타이드에 대해 100%이고 나머지에 대해서는 0% 만큼 낮다. 그와 같은 경우에, DNA-표적화 서열은 그 길이가 7 개의 뉴클레오타이드인 것으로 간주될 수 있다.
DNA - 표적화 RNA 의 단백질-결합 세그먼트
당해 DNA-표적화 RNA의 단백질-결합 세그먼트는 부위 지향적 변형 폴리펩타이드와 상호작용한다. 당해 DNA-표적화 RNA는 상기 언급된 DNA-표적화 분절를 통해 표적 DNA 내에서 특이적 뉴클레오타이드 서열에 대한 결합된 폴리펩타이드를 안내한다. 당해 DNA-표적화 RNA의 단백질-결합 세그먼트는 서로 상보적인 뉴클레오타이드의 2 개의 스트레치를 포함한다. 단백질-결합 세그먼트의 상보적 뉴클레오타이드는 혼성화되어 이중가닥 RNA 듀플렉스를 형성한다 (dsRNA) (참고 도 1a 및 1b).
당해 이중-분자 DNA-표적화 RNA는 2 개의 별개의 RNA 분자를 포함한다. 당해 이중-분자 DNA-표적화 RNA의각각의 2 개의 RNA 분자는 서로 상보적인 뉴클레오타이드의 스트레치를 포함하고, 이로써 2 개의 RNA 분자의 상보적 뉴클레오타이드 혼성화되어 단백질-결합 세그먼트의 이중가닥 RNA 듀플렉스를 형성한다 (도 1a).
일부 구현예에서, 활성제-RNA의 듀플렉스-형성 분절은 적어도 8 개의 인접 뉴클레오타이드의 스트레치에 대해 서열번호:431-562에서 제시된 활성제-RNA (tracrRNA) 분자 중의 하나, 또는 그의 보체와 적어도 약 60% 동일하다. 예를 들면, 활성제-RNA (또는 활성제-RNA의 듀플렉스-형성 분절을 인코딩하는 DNA) 의 듀플렉스-형성 분절은 적어도 8 개의 인접 뉴클레오타이드의 스트레치에 대해 서열번호:431-562에서 제시된 tracrRNA 서열 중의 하나, 또는 그의 보체와 적어도 약 60%, 적어도 약 65%, 적어도 약 70%, 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 98%, 적어도 약 99%, 또는 100 % 동일하다.
일부 구현예에서, 타겟터-RNA의 듀플렉스-형성 분절은 적어도 8 개의 인접 뉴클레오타이드의 스트레치에 대해 서열번호:563-679에서 제시된 타겟터-RNA (crRNA) 서열 중의 하나, 또는 그의 보체와적어도 약 60% 동일하다. 예를 들면, 타겟터-RNA (또는 타겟터-RNA의 듀플렉스-형성 분절을 인코딩하는 DNA) 의 듀플렉스-형성 분절은 적어도 8 개의 인접 뉴클레오타이드의 스트레치에 대해 서열번호:563-679에서 제시된 crRNA 서열 중의 하나, 또는 그의 보체와 적어도 약 65%, 적어도 약 70%, 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 98%, 적어도 약 99% 또는 100 % 동일하다.
2-분자 DNA-표적화 RNA는 타겟터-RNA와 활성제-RNA와의 조절된 (즉 조건부) 결합을 허용하도록 설계될 수 있다. 2-분자 DNA-표적화 RNA는, 활성제-RNA 및 타겟터-RNA 둘 모두가 dCas9와의 기능성 복합물에서 결합되지 않는다면 기능성이 없기 때문에, 2-분자 DNA-표적화 RNA는 활성제-RNA와 타겟터-RNA 사이의 결합이 유도성이 되도록 하여 유도성 (예를 들면, 약물 유도성)일 수 있다. 하나의 비제한적인 예로서, RNA 압타머는 활성제-RNA와 타겟터-RNA와의 결합을 조절 (즉, 제어)하기 위해 사용될 수 있다. 따라서, 활성제-RNA 및/또는 타겟터-RNA는 RNA 압타머 서열을 포함할 수 있다.
RNA 압타머는 대상체기술에 공지되어 있고 일반적으로 리보스위치의 합성 버전이다. 용어들 "RNA 압타머" 및 "리보스위치"는 일부인 RNA 분자의 구조의 유도성 조절 (및 따라서 특이적 서열의 이용가능성)을 제공하는 합성 및 천연 핵산 서열 둘 모두를 포함하도록 본원에서 상호교환적으로 사용된다. RNA 압타머는 보통 특정한 약물 (예를 들면, 소분자)에 특이적으로 결합하는 특정한 구조 (예를 들면, 헤어핀)로 접히는 서열을 포함한다. 약물의 결합은 압타머가 일부인 핵산의 특징을 변화시키는 RNA의 폴딩에서 구조적 변화를 야기한다. 비-제한적 예로서: (i) 압타머를 갖는 활성제-RNA는, 압타머가 적절한 약물에 의해 결합되지 않으면 동족 타겟터-RNA에 결합할 수 없다; (ii) 압타머를 갖는 타겟터-RNA는, 압타머가 적절한 약물에 의해 결합되지 않으면 동족 활성제-RNA에 결합할 수 없다; 및 (iii), 상이한 약물에 결합하는 상이한 압타머를 포함하는 타겟터-RNA 및 활성제-RNA 각각은, 약물 둘 모두가 존재하지 않으면 서로 결합할 수 없다. 이들 예에서 실증된 바와 같이, 2-분자 DNA-표적화 RNA는 유도성이도록 설계될 수 있다.
압타머 및 리보스위치의 예는, 예를 들면, 하기에서 발견될 수 있다: Nakamura 등, Genes Cells. 2012 May;17(5):344-64; Vavalle 등, Future Cardiol. 2012 May;8(3):371-82; Citartan 등, Biosens Bioelectron. 2012 Apr 15;34(1):1-11; 및 Liberman 등, Wiley Interdiscip Rev RNA. 2012 May-Jun;3(3):369-84; 이들 모두는 참조로 그 전체가 본원에 편입되어 있다.
2-분자 DNA-표적화 RNA에서 포함될 수 있는 뉴클레오타이드 서열의 비-제한적인 예는 서열번호:431-562에서 제시된 서열, 또는 그의 보체를 포함하고, 이들은 서열번호:563-679에서 제시된 임의의 서열, 또는 단백질 결합 세그먼트를 형성할 수 있는 그의 보체의 짝짓기를 한다.
당해 단일-분자 DNA-표적화 RNA는 뉴클레오타이드의 2 개의 스트레치 (타겟터-RNA 및 활성제-RNA)를 포함하고, 이들은 서로 상보적이고, 개재 뉴클레오타이드 ("링커" 또는 "링커 뉴클레오타이드")에 의해 공유 결합되고, 혼성화되어 단백질-결합 세그먼트의 이중가닥 RNA 듀플렉스 (dsRNA 듀플렉스)를 형성하고, 따라서 스템-루프 구조를 생기게 한다 (도 1b). 타겟터-RNA 및 활성제-RNA는 타겟터-RNA의 3' 말단 및 활성제-RNA의 5' 말단을 통해 공유결합될 수 있다. 대안적으로, 타겟터-RNA 및 활성제-RNA는 타겟터-RNA의 5' 말단 및 활성제-RNA의 3' 말단을 통해 공유 결합될 수 있다.
단일-분자 DNA-표적화 RNA의 링커는 그 길이가 약 3 뉴클레오타이드 내지 약 100 개의 뉴클레오타이드. 예를 들면, 링커는 그 길이가 약 3 뉴클레오타이드 (nt) 내지 약 90 nt, 약 3 뉴클레오타이드 (nt) 내지 약 80 nt, 약 3 뉴클레오타이드 (nt) 내지 약 70 nt, 약 3 뉴클레오타이드 (nt) 내지 약 60 nt, 약 3 뉴클레오타이드 (nt) 내지 약 50 nt, 약 3 뉴클레오타이드 (nt) 내지 약 40 nt, 약 3 뉴클레오타이드 (nt) 내지 약 30 nt, 약 3 뉴클레오타이드 (nt) 내지 약 20 nt 또는 약 3 뉴클레오타이드 (nt) 내지 약 10 nt일 수 있다. 예를 들면, 링커는 그 길이가 약 3 nt 내지 약 5 nt, 약 5 nt 내지 약 10 nt, 약 10 nt 내지 약 15 nt, 약 15 nt 내지 약 20 nt, 약 20 nt 내지 약 25 nt, 약 25 nt 내지 약 30 nt, 약 30 nt 내지 약 35 nt, 약 35 nt 내지 약 40 nt, 약 40 nt 내지 약 50 nt, 약 50 nt 내지 약 60 nt, 약 60 nt 내지 약 70 nt, 약 70 nt 내지 약 80 nt, 약 80 nt 내지 약 90 nt, 또는 약 90 nt 내지 약 100 nt일 수 있다. 일부 구현예에서, 단일-분자 DNA-표적화 RNA의 링커는 4 nt이다.
예시적인 단일-분자 DNA-표적화 RNA는 혼성화되어 dsRNA 듀플렉스를 형성하는 뉴클레오타이드의 2 개의 상보적 스트레치를 포함한다. 일부 구현예에서, 단일-분자 DNA-표적화 RNA (또는 스트레치를 인코딩하는 DNA)의 뉴클레오타이드의 2 개의 상보적 스트레치 중의 하나는 적어도 8 개의 인접 뉴클레오타이드의 스트레치에 대해 서열번호:431-562에서 제시된 활성제-RNA (tracrRNA) 분자 중의 하나, 또는 그의 보체 와 적어도 약 60% 동일하다. 예를 들면, 단일-분자 DNA-표적화 RNA (또는 스트레치를 인코딩하는 DNA)의 뉴클레오타이드의 2 개의 상보적 스트레치 중의 하나는 적어도 8 개의 인접 뉴클레오타이드의 스트레치에 대해 서열번호:431-562에서 제시된 tracrRNA 서열, 또는 그의 보체 중의 하나와 적어도 약 65%, 적어도 약 70%, 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 98%, 적어도 약 99% 또는 100 % 동일하다.
일부 구현예에서, 단일-분자 DNA-표적화 RNA (또는 스트레치를 인코딩하는 DNA)의 뉴클레오타이드의 2 개의 상보적 스트레치 중의 하나는 적어도 8 개의 인접 뉴클레오타이드의 스트레치에 대해 서열번호:563-679에서 제시된 타겟터-RNA (crRNA) 서열, 또는 그의 보체의 하나와 적어도 약 60% 동일하다. 예를 들면, 단일-분자 DNA-표적화 RNA (또는 스트레치를 인코딩하는 DNA)의 뉴클레오타이드의 2 개의 상보적 스트레치 중의 하나는 적어도 8 개의 인접 뉴클레오타이드의 스트레치에 대해 서열번호:563-679에서 제시된 crRNA 서열 중의 하나, 또는 그의 보체와 적어도 약 65%, 적어도 약 70%, 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 98%, 적어도 약 99% 동일한 또는 100 % 동일하다.
crRNA 및 tracrRNA의 적절한 자연 발생 동족 쌍은 적절한 동족 쌍을 결정할 때 (단백질-결합 도메인의 dsRNA 듀플렉스에 대한) 종 명칭 및 염기 짝짓기를 고려하여 서열번호:431-679에 대해 일상적으로 결정될 수 있다 (참고 비제한적인 예로서 도 8 참고).
당해 단일-분자 DNA-표적화 RNA 및 당해 이중-분자 DNA-표적화 RNA 둘 모두에 대해, 도 57은, tracrRNA 및 crRNA과 아주 조금(거의 50% 동일성) 공유하는 인공 서열이 DNA-표적화 RNA의 단백질-결합 도메인의 구조는 보존되는 한 표적 DNA를 절단하기 위해 Cas9와 함께 기능할 수 있다는 것을 설명한다. 따라서, DNA-표적화 RNA의 천연 발생 단백질-결합 도메인의 RNA 폴딩 구조는 인공 단백질-결합 도메인 (2-분자 또는 단일-분자 버전)을 설계 하기위해 고려될 수 있다. 비제한적인 예로서, 도 57의 기능성 인공 DNA-표적화 RNA는 (예를 들면, RNA 듀플렉스를 따라 동일한 수의 염기쌍을 포함하고 자연 발생 RNA에서 존재하는 바와 같은 동일한 "벌쥐(buldge)" 영역을 포함하는) 자연 발생 DNA-표적화의 단백질-결합 세그먼트의 구조를 기반으로 설계되었다. 구조는 임의의 종으로부터 임의의 자연 발생 crRNA:tracrRNA 쌍(다양한 종으로부터 crRNA 및 tracrRNA 서열에 대한 서열번호:431-679 참고)에 대한 당해분야의 숙련가에 의해 쉽게 생산될 수 있기 때문에, 인공 DNA-표적-RNA는 종으로부터 Cas9 (또는 관련된 Cas9, 참고 도 32a)를 사용할 때 주어진 종에 대한 천연 구조를 모방하도록 설계될 수 있다. (참고 도 24D 및 실시예 1에서 관련된 세부사항). 따라서, 적당한 DNA-표적화 RNA는 자연 발생 DNA-표적화 RNA의 단백질-결합 도메인의 구조를 모방하도록 설계된 단백질-결합 도메인을 포함하는 인공적으로 설계된 RNA (비-자연 발생)일 수 있다. (참고 서열번호:431-679, 적절한 동족 쌍을 측정할 때 종 명칭을 고려함).
단백질-결합 세그먼트는 그 길이가 약 10 개의 뉴클레오타이드 내지 약 100 개의 뉴클레오타이드일 수 있다. 예를 들면, 단백질-결합 세그먼트는 그 길이가 약 15 개의 뉴클레오타이드 (nt) 내지 약 80 nt, 약 15 nt 내지 약 50 nt, 약 15 nt 내지 약 40 nt, 약 15 nt 내지 약 30 nt 또는 약 15 nt 내지 약 25 nt일 수 있다.
당해 단일-분자 DNA-표적화 RNA 및 당해 이중-분자 DNA-표적화 RNA 둘 모두에 대해서도, 단백질-결합 세그먼트의 dsRNA 듀플렉스는 그 길이가 약 6 염기쌍s (bp) 내지 약 50bp일 수 있다. 예를 들면, 단백질-결합 세그먼트의 dsRNA 듀플렉스는 그 길이가 약 6 bp 내지 약 40 bp, 약 6 bp 내지 약 30bp, 약 6 bp 내지 약 25 bp, 약 6 bp 내지 약 20 bp, 약 6 bp 내지 약 15 bp, 약 8 bp 내지 약 40 bp, 약 8 bp 내지 약 30bp, 약 8 bp 내지 약 25 bp, 약 8 bp 내지 약 20 bp 또는 약 8 bp 내지 약 15 bp일 수 있다. 예를 들면, 단백질-결합 세그먼트의 dsRNA 듀플렉스는 그 길이가 약 약 8 bp 내지 약 10 bp, 약 10 bp 내지 약 15 bp, 약 15 bp 내지 약 18 bp, 약 18 bp 내지 약 20 bp, 약 20 bp 내지 약 25 bp, 약 25 bp 내지 약 30 bp, 약 30 bp 내지 약 35 bp, 약 35 bp 내지 약 40 bp, 또는 약 40 bp 내지 약 50 bp일 수 있다. 일부 구현예에서, 단백질-결합 세그먼트의 dsRNA 듀플렉스는 그 길이가 36 개의 염기쌍이다. 혼성화되어 단백질-결합 세그먼트의 dsRNA 듀플렉스를 형성하는 뉴클레오타이드 서열 사이의 상보성 퍼센트는 적어도 약 60%일 수 있다. 예를 들면, the 혼성화되어 단백질-결합 세그먼트의 dsRNA 듀플렉스를 형성하는 뉴클레오타이드 서열 사이의 상보성 퍼센트는 적어도 약 65%, 적어도 약 70%, 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 98%, 또는 적어도 약 99%일 수 있다. 일부 경우에서, 혼성화되어 단백질-결합 세그먼트의 dsRNA 듀플렉스를 형성하는 뉴클레오타이드 서열 사이의 상보성 퍼센트는 100%이다.
부위 지향적 변형 폴리펩타이드
당해 DNA-표적화 RNA 및 당해 부위 지향적 변형 폴리펩타이드는 복합물을 형성한다. DNA-표적화 RNA는 표적 DNA의 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하여 복합물에 대한 표적 특이성을 제공한다 (상기에서 언급 바와 같이). 복합물의 부위 지향적 변형 폴리펩타이드는 부위 특이적인 활성을 제공한다. 환언하면, 부위 지향적 변형 폴리펩타이드는 적어도 DNA-표적화 RNA의 단백질-결합 세그먼트와의 회합에 의해 DNA 서열 (예를 들면 염색체 서열 또는 염색체외 서열, 예를 들면 에피솜 서열, 미니서클 서열, 미토콘드리아 서열, 엽록체 서열, 등)로 유도된다 (상기에서 기재됨).
당해 부위 지향적 변형 폴리펩타이드는 표적 DNA (예를 들면, 표적 DNA의 절단 또는 메틸화) 및/또는 표적 DNA와 연관된 폴리펩타이드 (예를 들면, 히스톤 테일의 메틸화 또는 아세틸화)를 변형시킨다. 부위 지향적 변형 폴리펩타이드는 또한 "부위 지향적 폴리펩타이드"로서 본원에서 칭하거나 "RNA 결합 부위-유도된 변형 폴리펩타이드"로서 칭해진다.
일부 경우에서, 부위 지향적 변형 폴리펩타이드는 자연-발생 변형 폴리펩타이드이다. 다른 사례에서, 부위 지향적 변형 폴리펩타이드는 자연-발생 폴리펩타이드가 아니다 (예를 들면, 이하에서 논의된 바와 같은 키메라 폴리펩타이드 또는 변형된, 예를 들면, 돌연변이, 결실, 삽입된 자연-발생 폴리펩타이드).
예시적인 자연-발생 부위 지향적 변형 폴리펩타이드는 자연 발생 Cas9/Csn1 엔도뉴클레아제의 비제한적인 및 비-총망라한 목록으로서 서열번호:1-255에서 제시되어 있다. 이들 자연 발생 폴리펩타이드는, 본원에서 개시된 바와 같이, DNA-표적화 RNA에 결합하고, 그렇게 함으로써 표적 DNA 내의 특이적 서열로 지향되고, 표적 DNA를 절단하여 이중 가닥 절단을 산출한다. 당해 부위 지향적 변형 폴리펩타이드는 2 개의 부분, RNA-결합부 및 활성부를 포함한다. 일부 구현예에서, 당해 부위 지향적 변형 폴리펩타이드는 하기를 포함한다: (i) DNA-표적화 RNA와 상호작용하는 RNA-결합부로서, 상기 DNA-표적화 RNA는 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 결합부; 및 (ii) 부위 지향적 효소 활성을 나타내는 활성부 (예를 들면, DNA 메틸화를 위한 활성, DNA 절단을 위한 활성, 히스톤 아세틸화를 위한 활성, 히스톤 메틸화를 위한 활성, 등), 여기서 상기 효소 활성의 부위는 DNA-표적화 RNA에 의해 결정된다.
다른 구현예에서, 당해 부위 지향적 변형 폴리펩타이드는 하기를 포함한다: (i) DNA-표적화 RNA와 상호작용하는 RNA-결합부로서, 상기 DNA-표적화 RNA는 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 결합부; 및 (ii) 표적 DNA 내에서 전사를 조절하는 활성부 (예를 들면, 전사를 증가 또는 감소시키기 위함), 여기서 상기 표적 DNA 내의 조절된 전사의 부위는 DNA-표적화 RNA에 의해 결정된다.
일부 경우에서, 당해 부위 지향적 변형 폴리펩타이드는 표적 DNA를 변형시키는 효소 활성 (예를 들면, 뉴클레아제 활성, 메틸전이효소 활성, 탈메틸효소 활성, DNA 치유 활성, DNA 손상 활성, 탈아미노화 활성, 디스무타제 활성, 알킬화 활성, 탈퓨린화 활성, 산화 활성, 피리미딘 이량체 형성 활성, 인테그라제 활성, 전위효소 활성, 재조합효소 활성, 폴리머라제 활성, 리가제 활성, 헬리카제 활성, 광분해효소 활성 또는 글리코실라제 활성)을 갖는다.
다른 사례에서, 당해 부위 지향적 변형 폴리펩타이드는 표적 DNA와 연관된 폴리펩타이드 (예를 들면, 히스톤)를 변형시키는 효소 활성 (예를 들면, 메틸전이효소 활성, 탈메틸효소 활성, 아세틸전이효소 활성, 탈아세틸화효소 활성, 키나아제 활성, 포스파타제 활성, 우비퀴틴 리가제 활성, 탈우비퀴틴화 활성, 아데닐화 활성, 탈아데닐화 활성, 스모화 활성, 탈스모화 활성, 리보실화 활성, 탈리보실화 활성, 미리스토일화 활성 또는 탈미리스토일화 활성)을 갖는다.
예시적인 부위 지향적 변형 폴리펩타이드
일부 경우에서, 부위 지향적 변형 폴리펩타이드는 아미노산 서열을 포함하고, 이 서열은 도 3에서 묘사된 Cas9/Csn1 아미노산 서열의 아미노산 7-166 또는 731-1003과, 또는 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 상응하는 부분과 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 99%, 또는 100%, 아미노산 서열 동일성을 갖는다.
핵산 변형
일부 구현예에서, 당해 핵산 (예를 들면, DNA-표적화 RNA)은 핵산에 신규 또는 증대된 특징 (예를 들면, 개선된 안정성)을 제공하기 위해 1 이상의 변형, 예를 들면, 염기 변형, 골격 변형, 등을 포함한다. 대상체기술에 공지된 바와 같이, 뉴클레오사이드는 염기 -당 조합이다. 뉴클레오사이드의 염기부는 보통 헤테로사이클릭 염기이다. 그와 같은 헤테로사이클릭 염기의 2 개의 대부분의 공통의 클래스는 퓨린 및 피리미딘이다. 뉴클레오타이드는 뉴클레오사이드의 당부에 공유 결합된 포스페이트 그룹을 추가로 포함하는 뉴클레오사이드이다. 펜토푸라노실 당을 포함하는 뉴클레오사이드에 대해, 포스페이트 그룹은 당의 2', 3', 또는 5' 하이드록실 모이어티에 연결될 수 있다. 올리고뉴클레오타이드의 형성 시, 포스페이트 그룹은 인접한 뉴클레오사이드에 서로 공유결합하여 선형 폴리머 화합물을 형성한다. 결국, 이러한 선형 폴리머 화합물의 각 말단은 추가로 연결되어 원형 화합물을 형성할 수 있고, 그러나, 선형 화합물은 일반적으로 적당한아다. 또한, 선형 화합물은 내부 뉴클레오타이드 염기 상보성을 가질 수 있고 따라서 완전한 또는 부분적인 이중-가닥 화합물를 생산하기 위한 방식으로 폴딩될 수 있다. 올리고뉴클레오타이드 내에, 포스페이트 그룹은 통상적으로 올리고뉴클레오타이드의 뉴클레오사이드간 골격을 형성하는 것으로서 언급된다. RNA 및 DNA의 통상적인 연결 또는 골격은 3' 내지 5' 포스포디에스테르 연결이다.
변형된 골격 및 변형된 뉴클레오사이드간 연결
변형을 함유하는 적당한 핵산의 예는 변형된 골격 또는 비-천연 뉴클레오사이드간 연결을 함유하는 핵산을 포함한다. (변형된 골격을 갖는) 핵산은 골격에서 인 원자를 보유하는 것 및 골격에서 인 원자를 갖지 않는 것을 포함한다.
본원에서 인 원자를 함유하는 적당한 변형된 올리고뉴클레오타이드 골격은, 예를 들면, 포스포로티오에이트, 키랄 포스포로티오에이트, 포스포로디티오에이트, 포스포트리에스테르, 아미노알킬포스포트리에스테르, 메틸 및 3'-알킬렌 포스포네이트, 5'-알킬렌 포스포네이트 및 키랄 포스포네이트를 포함하는 다른 알킬 포스포네이트, 포스피네이트, 3'-아미노 포스포르아미데이트 및 아미노알킬포스포르아미데이트를 포함하는 포스포르아미데이트, 포스포로디아미데이트, 티오노포스포르아미데이트, 티오노알킬포스포네이트, 티오노알킬포스포트리에스테르, 노르말 3'-5' 연결을 갖는 셀레노포스페이트 및 포라노포스페이트, 이들의 2'-5' 연결된 유사체, 및 1 이상의 뉴클레오타이드간 연결이 3' 내지 3', 5' 내지 5' 또는 2' 내지 2' 연결인 역전된 극성을 갖는 것들을 포함한다. 역전된 극성을 갖는 적당한 올리고뉴클레오타이드는 3'-최대 뉴클레오타이드간 연결 즉 염기성일 수 있는 단일 역전된 뉴클레오사이드 잔기에서 단일 3' 내지 3' 연결을 포함한다 (핵염기는 누락되거나 그것 대신에 하이드록실 그룹을 갖는다). 다양한 염 (예컨대, 칼륨 또는 나트륨), 혼합된 염 및 유리 산 형태가 또한 포함된다.
일부 구현예에서, 당해 핵산은 하기를 포함한다: 1 이상의 포스포로티오에이트 및/또는 헤테로원자 뉴클레오사이드간 연결, 특히 -CH2-NH-O-CH2-, -CH2-N(CH3)-O-CH2- (메틸렌 (메틸이미노) 또는 MMI 골격으로서 공지됨), -CH2-O-N(CH3)-CH2-, -CH2-N(CH3)-N(CH3)-CH2- 및 -O-N(CH3)-CH2-CH2- (여기서 상기 원상태 포스포디에스테르 뉴클레오타이드간 연결은 -O-P(=O)(OH)-O-CH2-로서 나타낸다). MMI 유형 뉴클레오사이드간 연결은 상기 참조된 미국 특허 번호 5,489,677에서 개시되어 있다. 적당한 아마이드 뉴클레오사이드간 연결은 미국 특허 번호 5,602,240에서 개시되어 있다.
예를 들면, 미국 특허 번호 5,034,506에서 기재된 모폴리머 골격 구조를 갖는 핵산이 또한 적당하다. 예를 들면, 일부 구현예에서, 당해 핵산은 6-원 모폴리노 고리를 리보오스 고리 대신에 포함한다. 이들 구현예들 중 일부에서, 포스포로디아미데이트 또는 다른 비-포스포디에스테르 뉴클레오사이드간 연결은 포스포디에스테르 연결을 대체한다.
인 원자를 포함하지 않는 적당한 변형된 폴리뉴클레오타이드 골격은 단쇄 알킬 또는 사이클로알킬 뉴클레오사이드간 연결, 혼합된 헤테로원자 및 알킬 또는 사이클로알킬 뉴클레오사이드간 연결, 또는 1 이상의 단쇄 헤테로원자 또는 헤테로사이클릭 뉴클레오사이드간 연결에 의해 형성된 골격을 갖는다. 이들은 하기를 갖는 것들을 포함한다: 모폴리노 연결 (뉴클레오사이드의 당부로부터 부분적으로 형성됨); 실록산 골격; 설파이드, 설폭사이드 및 설폰 골격; 포름아세틸 및 티오포름아세틸 골격; 메틸렌 포름아세틸 및 티오포름아세틸 골격; 리보아세틸 골격; 알켄 함유 골격; 설파메이트 골격; 메틸렌이미노 및 메틸렌히드라지노 골격; 설포네이트 및 설폰아미드 골격; 아마이드 골격; 및 혼합된 N, O, S 및 CH2 성분부를 갖는 다른 것들.
모방체
당해 핵산은 핵산 모방체일 수 있다. 폴리뉴클레오타이드에 있는 그대로 적용된 용어 "모방체"는 폴리뉴클레오타이드를 포함하는 것으로 의도되고, 여기서 단지 푸라노스 고리 또는 푸라노스 고리 및 뉴클레오타이드간 연결 둘 모두는 비-푸라노스 그룹으로 대체되고, 단지 푸라노스 고리의 대체는 당 대용인 것으로 대상체기술에서 또한 언급된다. 헤테로사이클릭 염기 모이어티 또는 변형된 헤테로사이클릭 염기 모이어티는 적절한 표적 핵산과의 하이브리드화를 위해 유지된다. 탁월한 하이브리드화 특성을 갖는 것으로 보여진 하나의 그와 같은 핵산, 폴리뉴클레오타이드 모방체는 펩타이드 핵산 (PNA)로 불린다. PNA에서, 폴리뉴클레오타이드의 당-골격은 아마이드 함유 골격, 특히 아미노에틸글리신 골격으로 대체된다. 뉴클레오타이드는 유지되고 골격의 아마이드 부의 아자 질소 원자에 직접적으로 또는 간접적으로 결합된다.
탁월한 하이브리드화 특성을 갖는 것으로 보고된 하나의 폴리뉴클레오타이드 모방체는 펩타이드 핵산 (PNA)이다. PNA 화합물 중 골격은 2 이상의 연결된 아미노에틸글리신 단위이고, 이 단위는 PNA에 아마이드 함유 골격을 제공한다. 헤테로사이클릭 염기 모이어티는 골격의 아마이드부의 아자 질소 원자에 직접적으로 또는 간접적으로 결합된다. PNA 화합물의 제조를 기재하고 있는 대표적인 미국 특허들은, 비제한적으로 하기를 포함한다: 미국 특허 번호 5,539,082; 5,714,331; 및 5,719,262.
연구되었던 또 하나의 부류의 폴리뉴클레오타이드 모방체는 모폴리노 고리에 부착된 헤테로사이클릭 염기를 갖는 연결된 모폴리노 단위 (모폴리노 핵산)을 기반으로 한다. 모폴리노 핵산에서 모폴리노 모노머 단위를 연결하는 수많은 연결 그룹이 보고되었다. 하나의 부류의 연결 그룹은 비-이온성 올리고머 화합물을 제공하기 위해 선택되었다. 비-이온성 모폴리노-기반 올리고머 화합물은 세포성 단백질과의 원하지 않는 상호작용을 덜 가질 것 같다. 모폴리노-기반 폴리뉴클레오타이드는 세포성 단백질과의 원하지 않는 상호작용을 덜 형성할 것 같은 올리고뉴클레오타이드의 비-이온성 모사체이다 (Dwaine A. Braasch 및 David R. Corey, Biochemistry, 2002, 41(14), 4503-4510). 모폴리노-기반 폴리뉴클레오타이드는 미국 특허 번호 5,034,506에서 개시되어 있다. 모노머 서브유닛을 연결하는 다양한 상이한 연결 그룹을 갖는 모폴리노 부류의 폴리뉴클레오타이드 내의 다양한 화합물이 제조되어다.
추가 부류의 폴리뉴클레오타이드 모방체는 사이클로헥세닐 핵산 (CeNA)라고도 불린다. DNA/RNA 분자에서 통상적으로 존재하는 푸라노스 고리는 사이클로헥세닐 고리로 대체된다. CeNA DMT 보호된 포스포르아미다이트 단량체가 제조되었고 올리고머 화합물 합성 다음에 고전적 포스포르아미다이트 화학을 위해 사용되었다. 완전히 변형된 CeNA 올리고머 화합물 및 CeNA으로 변형된 특이적 위치를 갖는 올리고뉴클레오타이드가 제조 및 연구되었다 (참고 Wang 등, J. Am. Chem. Soc., 2000, 122, 8595-8602). 일반적으로 CeNA 단량체의 DNA 사슬로의 편입은 DNA/RNA 하이브리드의 안정성을 증가시킨다. CeNA 올리고아데닐레이트는 원상태 복합체에 대한 유사한 안정성을 갖는 RNA 및 DNA 보체와의 복합체를 형성했다. CeNA 구조를 천연 핵산 구조에 편입하는 연구는 용이한 형태적 적응을 계속하도록 NMR 및 원형 2색성에 의해 보여졌다.
추가 변형은 잠겨진 핵산 (LNA)를 포함하고, 여기서 2'-하이드록실 그룹은 당 고리의 4' 탄소 원자에 연결되고 그렇게 함으로써 2'-C,4'-C-옥시메틸렌 연결을 형성하고 그렇게 함으로써 바이사이클릭 당 모이어티를 형성한다. 연결은 2' 산소 원자 및 4' 탄소 원자를 브릿징하는 메틸렌 (-CH2-) 그룹일 수 있고, 여기서 n은 1 또는 2이다 (Singh 등, Chem. Commun., 1998, 4, 455-456). LNA 및 LNA 유사체는 상보적 DNA 및 RNA와의 매우 높은 듀플렉스 열 안정성 (Tm=+3 내지 +10 ℃), 3'-핵산외부분해에 대한 안정성 및 우수한 용해도 특성을 나타낸다. LNA를 함유하는 강력한 및 비독성 안티센스 올리고뉴클레오타이드는 기재되었다 (Wahlestedt 등, Proc. Natl. Acad. Sci. U.S.A., 2000, 97, 5633-5638).
그의 올리고머화, 및 핵산 인식 특성과 함께 LNA 단량체 아데닌, 시토신, 구아닌, 5-메틸-시토신, 티민 및 우라실의 합성 및 제조가 기재되었다 (Koshkin 등, Tetrahedron, 1998, 54, 3607-3630). LNA 및 이의 제조는 또한 WO 98/39352 및 WO 99/14226에서 기재되어 있다.
변형된 당 모이어티
당해 핵산은 1 이상의 치환된 당 모이어티를 또한 포함할 수 있다. 적당한 폴리뉴클레오타이드는 OH; F; O-, S-, 또는 N-알킬; O-, S-, 또는 N-알케닐; O-, S- 또는 N-알키닐; 또는 O-알킬-O-알킬로부터 선택된 당 치환기 그룹을 포함하고, 상기 알킬, 알케닐 및 알키닐은 치환된 또는 비치환된 C1 내지 C10 알킬 또는 C2 내지 C10 알케닐 및 알키닐일 수 있다. 특히 적당한 것은 O((CH2)nO) mCH3, O(CH2)nOCH3, O(CH2)nNH2, O(CH2)nCH3, O(CH2)nONH2, 및 O(CH2)nON((CH2)nCH3)2이고, 여기서 n 및 m은 1 내지 약 10이다. 다른 적당한 폴리뉴클레오타이드는 하기로부터 선택된 당 치환기 그룹을 포함한다: C1 내지 C10 저급 알킬, 치환된 저급 알킬, 알케닐, 알키닐, 알카릴, 아랄킬, O-알카릴 또는 O-아랄킬, SH, SCH3, OCN, Cl, Br, CN, CF3, OCF3, SOCH3, SO2CH3, ONO2, NO2, N3, NH2, 헤테로사이클로알킬, 헤테로사이클로알카릴, 아미노알킬아미노, 폴리알킬아미노, 치환된 실릴, RNA 절단 그룹, 리포터 그룹, 삽입제, 올리고뉴클레오타이드의 약력학적 특성을 형상시키는 그룹, 또는 올리고뉴클레오타이드의 약동학적 특성을 향상시키는 그룹, 및 유사한 특성을 갖는 다른 치환기. 적당한 변형은 2'-메톡시에톡시 (2'-O-CH2 CH2OCH3, 2'-O-(2-메톡시에틸) 또는 2'-MOE 로도 공지됨) (Martin 등, Helv. Chim. Acta, 1995, 78, 486-504) 즉, 알콕시알콕시 그룹을 포함한다. 추가 적당한 변형은 하기를 포함한다: 2'-디메틸아미노옥시에톡시, 즉, O(CH2)2ON(CH3)2 그룹, 이하의 실시예에서 기재됨 2'-DMAOE 로도 공지됨, 및 2'-디메틸아미노에톡시에톡시 (2'-O-디메틸-아미노-에톡시-에틸 또는 2'-DMAEOE로서 대상체기술에서도 공지됨), 즉, 2'-O-CH2-O-CH2-N(CH3)2.
다른 적당한 당 치환기 그룹은 메톡시 (-O-CH3), 아미노프로폭시 (--O CH2 CH2 CH2NH2), 알릴 (-CH2-CH=CH2), -O-알릴 (--O-- CH2-CH=CH2) 및 플루오로 (F)를 포함한다. 2'-당 치환기 그룹은 아라비노 (위) 위치 또는 리부 (아래) 위치에 있을 수 있다. 적당한 2'-아라비노 변형은 2'-F이다. 유사한 변형은 올리고머 화합물 상의 다른 위치, 특히 3' 말단 뉴클레오사이드 상의 또는 2'-5' 연결된 올리고뉴클레오타이드 내의 당의 3' 위치 및 5' 말단 뉴클레오타이드의 5' 위치에서 또한 만들어질 수 있다. 올리고머 화합물은 당 모방체 예컨대 사이클로부틸 모이어티를 펜토푸라노실 당 대신에 또한 가질 수 있다.
염기 변형 및 치환
당해 핵산은 핵염기 (종종 대상체기술에서 간단히 "염기"라고 불림) 변형 또는 치환을 또한 포함할 수 있다. 본원에서 사용된 바와 같이, "비변형된" 또는 "천연" 핵염기는 퓨린 염기 아데닌 (A) 및 구아닌 (G), 및 피리미딘 염기 티민 (T), 시토신 (C) 및 우라실 (U)을 포함한다. 변형된 핵염기는 하기를 포함한다: 다른 합성 및 천연 핵염기 예컨대 5-메틸시토신 (5-me-C), 5-하이드록시메틸 시토신, 잔틴, 하이포잔틴, 2-아미노아데닌, 아데닌 및 구아닌의 6-메틸 및 다른 알킬 유도체, 아데닌 및 구아닌의 2-프로필 및 다른 알킬 유도체, 2-티오우라실, 2-티오티민 및 2-티오시토신, 5-할로우라실 및 시토신, 5-프로피닐 (-C=C-CH3) 우라실 및 시토신 및 피리미딘 염기의 다른 알키닐 유도체, 6-아조 우라실, 시토신 및 티민, 5-우라실 (슈도우라실), 4-티오우라실, 8-할로, 8-아미노, 8-티올, 8-티오알킬, 8-하이드록실 및 다른 8-치환된 아데닌 및 구아닌, 5-할로 특히 5-브로모, 5-트리플루오로메틸 및 다른 5-치환된 우라실 및 시토신, 7-메틸구아닌 및 7-메틸아데닌, 2-F-아데닌, 2-아미노-아데닌, 8-아자구아닌 및 8-아자아데닌, 7-데아자구아닌 및 7-데아자아데닌 및 3-데아자구아닌 및 3-데아자아데닌. 추가의 변형된 핵염기는 하기를 포함한다: 트리사이클릭 피리미딘 예컨대 펜옥사진 시티딘(1H-피리미도(5,4-b)(1,4)벤족사진-2(3H)-온), 페노티아진 시티딘 (1H-피리미도(5,4-b)(1,4)벤조티아진-2(3H)-온), G-c램프 예컨대 치환된 펜옥사진 시티딘 (예를 들면 9-(2-아미노에톡시)-H-피리미도(5,4-(b) (1,4)벤족사진-2(3H)-온), 카바졸 시티딘 (2H-피리미도(4,5-b)인돌-2-온), 피리도인돌 시티딘 (H-피리도(3',2':4,5)피롤로(2,3-d)피리미딘-2-온).
헤테로사이클릭 염기 모이어티는, 퓨린 또는 피리미딘 염기가 다른 헤테로사이클, 예를 들면 7-데아자-아데닌, 7-데아자구아노신, 2-아미노피리딘 및 2-피리돈으로 치환된 것을 또한 포함할 수 있다. 추가의 핵염기는 하기를 포함한다: 미국 특허 번호 3,687,808에서 개시된 것들, The Concise Encyclopedia Of Polymer Science And Engineering, 페이지 858-859, Kroschwitz, J. I., ed. John Wiley & Sons, 1990에서 개시된 것들, Englisch 등, Angewandte Chemie, International Edition, 1991, 30, 613에 의해 개시된 것들, 및 Sanghvi, Y. S., Chapter 15, Antisense Research and Applications, 페이지 289-302, Crooke, S. T. 및 Lebleu, B., ed., CRC Press, 1993에 의해 개시된 것들. 이들 핵염기의 어떤 것은 올리고머 화합물의 결합 친화도를 증가시키는데 유용하다. 이들은 5-치환된 피리미딘, 6-아자피리미딘 및 N-2, N-6 및 O-6 치환된 퓨린을 포함하고, 이들은 2-아미노프로필아데닌, 5-프로피닐우라실 및 5-프로피닐시토신을 포함한다. 5-메틸시토신 치환은 0.6-1.2 ℃ (Sanghvi 등, eds., Antisense Research and Applications, CRC Press, Boca Raton, 1993, pp. 276-278)에 의해 핵산 듀플렉스 안정성을 증가시키는 것으로 보여졌고 예를 들면, 2'-O-메톡시에틸 당 변형와 조합될 때, 적당한 염기 치환이다.
콘주게이트
당해 핵산의 또 하나의 가능한 변형은 폴리뉴클레오타이드에, 올리고뉴클레오타이드의 활성, 세포성 분포 또는 세포성 흡수를 향상시키는 1 이상의 모이어티 또는 콘주게이트를 화학적으로 연결하는 것을 수반한다. 이들 모이어티 또는 콘주게이트는 관능 그룹 예컨대 일차 또는 2차 하이드록실 그룹에 공유결합된 콘주게이트를 포함할 수 있다. 콘주게이트 그룹은, 비제한적으로, 삽입제, 리포터 분자, 폴리아민, 폴리아미드, 폴리에틸렌 글리콜, 폴리에테르, 올리고머의 약동학적 특성을 향상시키는 그룹, 및 올리고머의 약력학적 특성을 향상시키는 그룹을 포함한다. 적당한 콘주게이트 그룹은, 비제한적으로, 콜레스테롤, 지질, 인지질, 바이오틴, 펜아진, 폴레이트, 펜안트리딘, 안트라퀴논, 아크리딘, 플루오레신, 로다민, 쿠마린, 및 염료를 포함한다. 약동학적 특성을 향상시키는 그룹은 흡수를 개선하는 그룹, 분해에 대한 내성을 향상시키고/거나, 표적 핵산과의 서열-특이적 하이브리드화를 강화시키는 그룹을 포함한다. 약력학적 특성을 향상시키는 그룹은 당해 핵산의 흡수, 분포, 대사 또는 배출을 개선하는 그룹을 포함한다.
콘주게이트 모이어티는 비제한적으로 하기를 포함한다: 지질 모이어티 예컨대 콜레스테롤 모이어티 (Letsinger 등, Proc. Natl. Acad. Sci. USA, 1989, 86, 6553-6556), 콜산 (Manoharan 등, Bioorg. Med. Chem. Let., 1994, 4, 1053-1060), 티오에테르, 예를 들면, 헥실-S-트리틸티올 (Manoharan 등, Ann. N.Y. Acad. Sci., 1992, 660, 306-309; Manoharan 등, Bioorg. Med. Chem. Let., 1993, 3, 2765-2770), 티오콜레스테롤 (Oberhauser 등, Nucl. Acids Res., 1992, 20, 533-538), 지방족 사슬, 예를 들면, 도데칸디올 또는 운데실 잔기 (Saison-Behmoaras 등, EMBO J., 1991, 10, 1111-1118; Kabanov 등, FEBS Lett., 1990, 259, 327-330; Svinarchuk 등, Biochimie, 1993, 75, 49-54), 인지질, 예를 들면, 디-헥사데실-rac-글리세롤 또는 트리에틸암모늄 1,2-디-O-헥사데실-rac-글리세로-3-H-포스포네이트 (Manoharan 등, Tetrahedron Lett., 1995, 36, 3651-3654; Shea 등, Nucl. Acids Res., 1990, 18, 3777-3783), 폴리아민 또는 폴리에틸렌 글리콜 사슬 (Manoharan 등, Nucleosides & Nucleotides, 1995, 14, 969-973), 또는 아다만탄 아세트산 (Manoharan 등, Tetrahedron Lett., 1995, 36, 3651-3654), 팔미틸 모이어티 (Mishra 등, Biochim. Biophys. Acta, 1995, 1264, 229-237), 또는 옥타데실아민 또는 헥실아미노-카보닐-옥시콜레스테롤 모이어티 (Crooke 등, J. Pharmacol. Exp. Ther., 1996, 277, 923-937.\
콘주게이트는 "단백질 형질도입 도메인" 또는 PTD (CPP - 세포 침투 펩타이드로도 공지됨)을 포함할 수 있고, 이것은 폴리펩타이드, 폴리뉴클레오타이드, 탄수화물, 또는 지질 이중층, 교질입자, 세포막, 소기관 막, 또는 소포 막을 가로지르는 것을 촉진하는 유기 또는 무기 화합물을 의미할 수 있다. 작은 극성 분자로부터 큰 거대분자 및/또는 나노입자 범위일 수 있는 또 하나의 분자에 부착된 PTD는, 막을 가로지르는, 예를 들면 세포외 공간으로부터 세포내 공간으로 이동하는 분자, 또는 소기관 내의 사이토졸을 촉진한다. 일부 구현예에서, PTD는 외인성 폴리펩타이드 (예를 들면, 부위 지향적 변형 폴리펩타이드)의 아미노 말단에 공유결합된다. 일부 구현예에서, PTD는 외인성 폴리펩타이드 (예를 들면, 부위 지향적 변형 폴리펩타이드)의 카복실 말단에 공유 결합된다. 일부 구현예에서, PTD는 핵산 (예를 들면, DNA-표적화 RNA, DNA-표적화 RNA를 인코딩하는 폴리뉴클레오타이드, 부위 지향적 변형 폴리펩타이드를 인코딩하는 폴리뉴클레오타이드, 등)에 공유결합된다. 예시적인 PTD는 비제한적으로 하기를 포함한다: 최소 운데카펩타이드 단백질 형질도입 도메인 (YGRKKRRQRRR; 서열번호:264를 포함하는 HIV-1 TAT의 잔기 47-57에 상응함); 세포에의 직접적인 유입에 충분한 수많은 아르기닌 (예를 들면, 3, 4, 5, 6, 7, 8, 9, 10, 또는 10-50 아르기닌)을 포함하는 폴리아르기닌 서열; VP22 도메인 (Zender 등 (2002)는 cancer Gene Ther. 9(6):489-96); 드로소필라 안네나페디아 단백질 형질도입 도메인 (Noguchi 등 (2003) Diabetes 52(7):1732-1737); 끝이 잘린 인간 칼시토닌 펩타이드 (Trehin 등 (2004) Pharm . Research 21:1248-1256); 폴리라이신 (Wender 등 (2000) Proc . Natl . Acad . Sci . USA 97:13003-13008); RRQRRTSKLMKR (서열번호:265); 트랜스포르탄 GWTLNSAGYLLGKINLKALAALAKKIL (서열번호:266); KALAWEAKLAKALAKALAKHLAKALAKALKCEA (서열번호:267); 및 RQIKIWFQNRRMKWKK (서열번호:268). 예시적인 PTD는 비제한적으로, YGRKKRRQRRR (서열번호:264), RKKRRQRRR (서열번호:269); 3 개의 아르기닌 잔기 내지 50 개의 아르기닌 잔기의 아르기닌 호모폴리머; 예시적인 PTD 도메인 아미노산 서열은, 비제한적으로, 하기의 임의의 것을 포함한다: YGRKKRRQRRR (서열번호:264); RKKRRQRR (서열번호:270); YARAAARQARA (서열번호:271); THRLPRRRRRR (서열번호:272); 및 GGRRARRRRRR (서열번호:273). 일부 구현예에서, PTD는 활성가능 CPP (ACPP)이다 (Aguilera 등 (2009) Integr Biol (Camb) June; 1(5-6): 371-381). ACPP는 순전하를 거의 제로로 감소시키고 그렇게 함으로써 세포에의 부착 및 흡수를 억제하는 일치되는 다중음이온 (예를 들면, Glu9 또는 "E9")에 대한 절단가능 링커를 통해 연결된 다중양이온성 CPP (예를 들면, Arg9 또는 "R9")를 포함한다. 링커의 절단시, 다중음이온가 방출되고, 폴리아르기닌 및 그의 고유한 부착성을 국소로 벗기고, 따라서 막을 가로지르기 위해 ACPP를 "활성화한다 ".
예시적인 DNA - 표적화 RNA
일부 구현예에서, 적당한 DNA-표적화 RNA는 2 개의 별개의 RNA 폴리뉴클레오타이드 분자를 포함한다. 2 개의 별개의 RNA 폴리뉴클레오타이드 분자 (활성제-RNA)의 첫 번째 것은 적어도 8 개의 인접 뉴클레오타이드의 스트레치에 대해 서열번호:431-562에서 제시된 뉴클레오타이드 서열의 임의의 하나와 적어도 약 60%, 적어도 약 65%, 적어도 약 70%, 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 98%, 적어도 약 99%, 또는 100% 뉴클레오타이드 서열 동일성을 갖는 뉴클레오타이드 서열또는 그의 보체를 포함한다. 2 개의 별개의 RNA 폴리뉴클레오타이드 분자 (타겟터-RNA)의 두 번째 것은 적어도 8 개의 인접 뉴클레오타이드의 스트레치에 대해 서열번호:563-679에서 제시된 뉴클레오타이드 서열의 임의의 하나, 또는 그의 보체와 적어도 약 60%, 적어도 약 65%, 적어도 약 70%, 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 98%, 적어도 약 99%, 또는 100% 뉴클레오타이드 서열 동일성을 갖는 뉴클레오타이드 서열을 포함한다.
일부 구현예에서, 적당한 DNA-표적화 RNA는 단일 RNA 폴리뉴클레오타이드를 포함하고 적어도 8 개의 인접 뉴클레오타이드의 스트레치에 대해 서열번호:431-562에서 제시된 뉴클레오타이드 서열의 임의의 하나와 적어도 약 60%, 적어도 약 65%, 적어도 약 70%, 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 98%, 적어도 약 99%, 또는 100% 뉴클레오타이드 서열 동일성을 갖는 제 1 뉴클레오타이드 서열 및 적어도 8 개의 인접 뉴클레오타이드의 스트레치에 대해 서열번호: 463-679에서 제시된 뉴클레오타이드 서열의 임의의 하나와 적어도 약 60%, 적어도 약 65%, 적어도 약 70%, 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 98%, 적어도 약 99%, 또는 100% 뉴클레오타이드 서열 동일성을 갖는 제 2 뉴클레오타이드 서열을 포함한다.
일부 구현예에서, DNA-표적화 RNA는 이중-분자 DNA-표적화 RNA를 포함하고 타겟터-RNA는 5' 말단에서 표적 DNA에 대해 상보적인 뉴클레오타이드의 스트레치에 연결된 서열 5'GUUUUAGAGCUA-3' (서열번호:679)을 포함한다. 일부 구현예에서, DNA-표적화 RNA는 이중-분자 DNA-표적화 RNA를 포함하고 활성제-RNA는 서열 5' UAGCAAGUUAAAAUAAGGCUAGUCCG-3' (서열번호://)를 포함한다.
일부 구현예에서, DNA-표적화 RNA는 단일-분자 DNA-표적화 RNA이고 5' 말단에서 표적 DNA에 대해 상보적인 뉴클레오타이드의 스트레치에 연결된 서열 5'-GUUUUAGAGCUA-링커-UAGCAAGUUAAAAUAAGGCUAGUCCG-3'를 포함한다 (여기서 "링커"는 임의의 뉴클레오타이드 서열을 포함할 수 있는 임의의 링커 뉴클레오타이드 서열을 나타낸다) (서열번호://). 다른 예시적인 단일-분자 DNA-표적화 RNA는 서열번호: 680-682에서 제시된 것을 포함한다.
당해 DNA - 표적화 RNA 및/또는 당해 부위 지향적 변형 폴리펩타이드를 인코딩하는 핵산
당해 개시내용은 당해 DNA-표적화 RNA 및/또는 당해 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산을 제공한다. 일부 구현예에서, 당해 DNA-표적화 RNA-인코딩 핵산은 발현 벡터, 예를 들면, 재조합 발현 벡터이다.
일부 구현예에서, 당해 방법은 표적 DNA를 접촉시키거나 세포 (또는 세포의 모집단)에 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 1 이상의 핵산에 도입하는 것을 수반한다. 일부 구현예에서 표적 DNA를 포함하는 세포는 시험관내에 있다. 일부 구현예에서 표적 DNA를 포함하는 세포는 생체내에 있다. DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 적당한 핵산은 발현 벡터를 포함하고, 여기서 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 발현 벡터는 "재조합 발현 벡터"이다.
일부 구현예에서, 재조합 발현 벡터는 바이러스 구조체, 예를 들면, 재조합 아데노-연관된 바이러스 구조체 (참고, 예를 들면, 미국 특허 번호 7,078,387), 재조합 아데노바이러스 구조체, 재조합 렌티바이러스 구조체, 재조합 레트로바이러스 구조체 등이다.
적당한 발현 벡터는, 비제한적으로 하기를 포함한다: 바이러스 벡터 (예를 들면 우두 바이러스를 기반으로 하는 바이러스 벡터; 폴리오바이러스; 아데노바이러스 (참고, 예를 들면, Li 등, Invest Opthalmol Vis Sci 35:2543 2549, 1994; Borras 등, Gene Ther 6:515 524, 1999; Li 및 Davidson, PNAS 92:7700 7704, 1995; Sakamoto 등, H Gene Ther 5:1088 1097, 1999; WO 94/12649, WO 93/03769; WO 93/19191; WO 94/28938; WO 95/11984 및 WO 95/00655); 아데노-연관된 바이러스 (참고, 예를 들면, Ali 등, Hum Gene Ther 9:81 86, 1998, Flannery 등, PNAS 94:6916 6921, 1997; Bennett 등, Invest Opthalmol Vis Sci 38:2857 2863, 1997; Jomary 등, Gene Ther 4:683 690, 1997, Rolling 등, Hum Gene Ther 10:641 648, 1999; Ali 등, Hum Mol Genet 5:591 594, 1996; Srivastava in WO 93/09239, Samulski 등, J. Vir. (1989) 63:3822-3828; Mendelson 등, Virol. (1988) 166:154-165; 및 Flotte 등, PNAS (1993) 90:10613-10617); SV40; 단순 포진 바이러스; 인간 면역결핍 바이러스 (참고, 예를 들면, Miyoshi 등, PNAS 94:10319 23, 1997; Takahashi 등, J Virol 73:7812 7816, 1999); 레트로바이러스 벡터 (예를 들면, 쥣과 백혈병 바이러스, 비장 괴사 바이러스, 및 레트로바이러스 예컨대 루(Rous) 육종 바이러스, 하비 육종 바이러스, 조류 백혈증 바이러스, 렌티바이러스, 인간 면역결핍 바이러스, 골수증식성 육종 바이러스, 및 유선 종양 바이러스로부터 유도된 벡터); 등.
수많은 적당한 발현 벡터는 당해분야의 숙련가에게 공지되어 있고, 많은 것들은 상업적으로 이용가능하다. 하기의 벡터는 진핵 숙주 세포: pXT1, pSG5 (Stratagene), pSVK3, pBPV, pMSG, 및 pSVLSV40 (Pharmacia)에 대해 예로서 제공된다. 그러나, 임의의 다른 벡터는 숙주세포와 있는 그대로 양립가능한 한 사용될 수 있다.
이용된 숙주/벡터 시스템에 따라, 항시적 및 유도성 프로모터, 전사 인핸서 요소, 전사 종결자 등을 포함하는 임의의 수의 적당한 전사 및 번역 조절 요소는 발현 벡터에서 사용될 수 있다 (참고 예를 들면, Bitter 등 (1987) Methods in Enzymology, 153:516-544).
일부 구현예에서, DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열은 조절 요소, 예를 들면, 전사 조절 요소, 예컨대 프로모터에 작동가능하게 연결된다. 전사 조절 요소는 진핵 세포, 예를 들면, 포유동물 세포; 또는 원핵 세포 (예를 들면, 박테리아 또는 고세균 세포)에서 기능적일 수 있다. 일부 구현예에서, DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열은 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열의 발현을 원핵 및 진핵 세포 둘 모두에서 허용하는 다중 조절 요소에 작동가능하게 연결된다.
적당한 진핵 프로모터 (진핵 세포에서 기능적인 프로모터)의 비-제한적인 예는 사이토메갈로바이러스 (CMV) 전초기, 단순 포진 바이러스 (HSV) 티미딘 키나아제, 초기 및 후기 SV40, 레트로바이러스로부터의 긴 말단 반복 (LTR), 및 마우스 메탈로티오네인-I로부터의 것들을 포함한다. 적절한 벡터 및 프로모터의 선택은 당업자의 수준 내에 있다. 발현 벡터는 번역 개시 및 전사 종결자를 위한 리보솜 결합 부위를 또한 함유할 수 있다. 발현 벡터는 발현을 증폭시키기 위한 적절한 서열을 또한 포함할 수 있다. 발현 벡터는 부위 지향적 변형 폴리펩타이드에 융합되고 따라서 키메라 폴리펩타이드를 생산하는 단백질 태그 (예를 들면, 6xHis 태그, 혈구응집소 태그, 녹색 형광 단백질, 등)을 인코딩하는 뉴클레오타이드 서열을 또한 포함할 수 있다.
일부 구현예에서, DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열은 유도성 프로모터에 작동가능하게 연결된다. 일부 구현예에서, DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열은 항시적 프로모터에 작동가능하게 연결된다.
핵산을 숙주세포에 도입하는 방법은 대상체기술에 공지되어 있고, 임의의 공지된 방법은 핵산 (예를 들면, 발현 구조체)을 세포에 도입하기 위해 사용될 수 있다. 적당한 방법은 예를 들면, 하기를 포함한다: 바이러스 또는 박테리오파아지 감염, 형질감염, 콘주게이션, 원형질 융합, 리포펙션, 전기천공, 칼슘 포스페이트 침전, 폴리에틸렌이민 (PEI)-매개된 형질감염, DEAE-덱스트란 매개된 형질감염, 리포좀-매개된 형질감염, 입자총 기술, 칼슘 포스페이트 침전, 직접적인 현미 주입, 나노입자-매개된 핵산 전달 (참고, 예를 들면, Panyam 등, Adv Drug Deliv Rev. 2012 Sep 13. pii: S0169-409X(12)00283-9. doi: 10.1016/j.addr.2012.09.023), 등.
키메라 폴리펩타이드
당해 개시내용은 키메라 부위 지향적 변형 폴리펩타이드를 제공한다. 당해 키메라 부위 지향적 변형 폴리펩타이드 당해 DNA-표적화 RNA (상기에서 기재됨)와 상호작용하고 (예를 들면, 그것에 결합한다). DNA-표적화 RNA는 키메라 부위 지향적 변형 폴리펩타이드를 표적 DNA 내의 표적 서열 (예를 들면 염색체 서열 또는 염색체외 서열, 예를 들면 에피솜 서열, 미니서클 서열, 미토콘드리아 서열, 엽록체 서열, 등)로 안내한다. 당해 키메라 부위 지향적 변형 폴리펩타이드는 표적 DNA (예를 들면, 표적 DNA의 절단 또는 메틸화) 및/또는 표적 DNA와 연관된 폴리펩타이드 (예를 들면, 히스톤 테일의 메틸화 또는 아세틸화)를 변형시킨다.
당해 키메라 부위 지향적 변형 폴리펩타이드는 표적 DNA (예를 들면, 표적 DNA의 절단 또는 메틸화) 및/또는 표적 DNA와 연관된 폴리펩타이드 (예를 들면, 히스톤 테일의 메틸화 또는 아세틸화)를 변형시킨다. 키메라 부위 지향적 변형 폴리펩타이드는 "키메라 부위 지향적 폴리펩타이드" 또는 "키메라 RNA 결합 부위-유도된 변형 폴리펩타이드"로도 본원에서 칭한다.
당해 키메라 부위 지향적 변형 폴리펩타이드는 2 개의 부분, RNA-결합부 및 활성부를 포함한다. 당해 키메라 부위 지향적 변형 폴리펩타이드는 적어도 2 개의 상이한 폴리펩타이드로부터 유도된 아미노산 서열을 포함한다. 당해 키메라 부위 지향적 변형 폴리펩타이드는 변형된 및/또는 자연-발생 폴리펩타이드 서열 (예를 들면, 변형된 또는 비변형된 Cas9/Csn1 단백질로부터의 제 1 아미노산 서열; 및 Cas9/Csn1 단백질 이외의 제 2 아미노산 서열)을 포함할 수 있다.
RNA -결합부
일부 경우에서, 당해 키메라 부위 지향적 변형 폴리펩타이드의 RNA-결합부는 자연-발생 폴리펩타이드이다. 다른 사례에서, 당해 키메라 부위 지향적 변형 폴리펩타이드의 RNA-결합부는 자연-발생 분자가 아니다 (변형된, 예를 들면, 돌연변이, 결실, 삽입). 관심 자연-발생 RNA-결합부는 대상체기술에서 공지된 부위 지향적 변형 폴리펩타이드로부터 유도된다. 예를 들면, 서열번호:1-256 및 795-1346은 부위 지향적 변형 폴리펩타이드로서 사용될 수 있는 자연 발생 Cas9/Csn1 엔도뉴클레아제의 비제한적인 및 비-총망라한 목록을 제공한다. 일부 경우에서, 당해 키메라 부위 지향적 변형 폴리펩타이드의 RNA-결합부는 아미노산 서열을 포함하고, 이 서열은 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것을 갖는 폴리펩타이드의 RNA-결합부에 대해 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 98%, 적어도 약 99%, 또는 100%, 아미노산 서열 동일성을 갖는다).
일부 경우에서, 부위 지향적 변형 폴리펩타이드는 아미노산 서열을 포함하고, 이 서열은 도 3에서 묘사된 Cas9/Csn1 아미노산 서열의 아미노산 7-166 또는 731-1003과, 또는 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 상응하는 부분과 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 99%, 또는 100%, 아미노산 서열 동일성을 갖는다.
활성부
RNA-결합부에 추가하여, 키메라 부위 지향적 변형 폴리펩타이드는 "활성부"를 포함한다. 일부 구현예에서, 당해 키메라 부위 지향적 변형 폴리펩타이드의 활성부는 부위 지향적 변형 폴리펩타이드의 자연-발생 활성부 (예를 들면, Cas9/Csn1 엔도뉴클레아제)를 포함한다. 다른 구현예에서, 당해 키메라 부위 지향적 변형 폴리펩타이드의 활성부는 부위 지향적 변형 폴리펩타이드의 자연-발생 활성부의 변형된 아미노산 서열 (예를 들면, 치환, 결실, 삽입)을 포함한다. 관심 자연-발생 활성부는 대상체기술에서 공지된 부위 지향적 변형 폴리펩타이드로부터 유도된다. 예를 들면, 서열번호:1-256 및 795-1346은 부위 지향적 변형 폴리펩타이드로서 사용될 수 있는 자연 발생 Cas9/Csn1 엔도뉴클레아제의 비제한적인 및 비-총망라한 목록을 제공한다. 당해 키메라 부위 지향적 변형 폴리펩타이드의 활성부는 가변이고 본원에서 개시된 방법에서 유용할 수 있는 임의의 이종기원 폴리펩타이드 서열을 포함할 수 있다.
일부 구현예에서, 당해 키메라 부위 지향적 변형 폴리펩타이드는 하기를 포함한다: (i) DNA-표적화 RNA와 상호작용하는 RNA-결합부로서, 상기 DNA-표적화 RNA는 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 결합부; 및 (ii) 부위 지향적 효소 활성을 나타내는 활성부 (예를 들면, DNA 메틸화를 위한 활성, DNA 절단을 위한 활성, 히스톤 아세틸화를 위한 활성, 히스톤 메틸화를 위한 활성, 등), 여기서 상기 효소 활성의 부위는 DNA-표적화 RNA에 의해 결정된다.
다른 구현예에서, 당해 키메라 부위 지향적 변형 폴리펩타이드는 하기를 포함한다: (i) DNA-표적화 RNA와 상호작용하는 RNA-결합부로서, 상기 DNA-표적화 RNA는 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 결합부; 및 (ii) 표적 DNA 내에서 전사를 조절하는 활성부 (예를 들면, 전사를 증가 또는 감소시키기 위함), 여기서 상기 표적 DNA 내의 조절된 전사의 부위는 DNA-표적화 RNA에 의해 결정된다.
일부 경우에서, 당해 키메라 부위 지향적 변형 폴리펩타이드의 활성부는 하기를 갖는다: 표적 DNA를 변형시키는 효소 활성 (예를 들면, 뉴클레아제 활성, 메틸전이효소 활성, 탈메틸효소 활성, DNA 치유 활성, DNA 손상 활성, 탈아미노화 활성, 디스무타제 활성, 알킬화 활성, 탈퓨린화 활성, 산화 활성, 피리미딘 이량체 형성 활성, 인테그라제 활성, 전위효소 활성, 재조합효소 활성, 폴리머라제 활성, 리가제 활성, 헬리카제 활성, 광분해효소 활성 또는 글리코실라제 활성).
다른 사례에서, 당해 키메라 부위 지향적 변형 폴리펩타이드의 활성부는 표적 DNA와 연관된 폴리펩타이드 (예를 들면, 히스톤)를 변형시키는 효소 활성 (예를 들면, 메틸전이효소 활성, 탈메틸효소 활성, 아세틸전이효소 활성, 탈아세틸화효소 활성, 키나아제 활성, 포스파타제 활성, 우비퀴틴 리가제 활성, 탈우비퀴틴화 활성, 아데닐화 활성, 탈아데닐화 활성, 스모화 활성, 탈스모화 활성, 리보실화 활성, 탈리보실화 활성, 미리스토일화 활성 또는 탈미리스토일화 활성)을 갖는다.
일부 경우에서, 당해 키메라 부위 지향적 변형 폴리펩타이드의 활성부는 효소 활성 (상기에서 기재됨)를 나타낸다. 다른 사례에서, 당해 키메라 부위 지향적 변형 폴리펩타이드의 활성부는 표적 DNA의 전사 (상기에서 기재됨)를 조절한다. 당해 키메라 부위 지향적 변형 폴리펩타이드의 활성부는 가변이고 본원에서 개시된 방법에서 유용할 수 있는 임의의 이종기원 폴리펩타이드 서열을 포함할 수 있다.
예시적인 키메라 부위 지향적 변형 폴리펩타이드
일부 구현예에서, 키메라 부위 지향적 변형 폴리펩타이드의 활성부는 변형된 형태의 Cas9/Csn1 단백질을 포함한다. 일부 예에서, 변형된 형태의 Cas9/Csn1 단백질은 아미노산 변화 (예를 들면, 결실, 삽입, 또는 치환)을 포함하고, 이 변화는 Cas9/Csn1 단백질의 자연-발생 뉴클레아제 활성을 감소시킨다. 예를 들면, 일부 예에서, 변형된 형태의 Cas9/Csn1 단백질은 상응하는 야생형 Cas9/Csn1 폴리펩타이드의 뉴클레아제 활성의 50% 미만, 40% 미만, 30% 미만, 20% 미만, 10% 미만, 5% 미만, 또는 1% 미만을 갖는다. 일부 경우에서, 변형된 형태의 Cas9/Csn1 폴리펩타이드는 실질적인 뉴클레아제 활성을 갖지 않는다.
일부 구현예에서, 변형된 형태의 Cas9/Csn1 폴리펩타이드는 D10A (서열번호:8의 아미노산 위치 10에서 아스파르테이트 내지 알라닌) 돌연변이 (또는 서열번호:1-256 및 795-1346에서 제공되는 임의의 단백질의 상응하는 돌연변이)이고, 이것은 표적 DNA의 상보적 가닥을 절단할 수 있지만 비-표적화 DNA의 상보적 가닥을 절단하기 위한 감소된 능력을 갖는다 (참고 도 11). 일부 구현예에서, 변형된 형태의 Cas9/Csn1 폴리펩타이드는 H840A (아미노산 위치 840에서 히스티딘 내지 알라닌) 돌연변이 (또는 서열번호:1-256 및 795-1346에서 제시된 임의의 단백질의 상응하는 돌연변이)이고, 이것은 비-표적화 DNA의 상보적 가닥을 절단할 수 있지만 표적 DNA의 상보적 가닥을 절단하기 위한 감소된 능력을 갖는다 (참고 도 11). 일부 구현예에서, 변형된 형태의 Cas9/Csn1 폴리펩타이드는 D10A 및 H840A 돌연변이 (또는 서열번호:1-256 및 795-1346에서 제시된 임의의 단백질의 상응하는 돌연변이)를 제공하고 이로써 폴리펩타이드는 표적화 DNA의 상보적 및 비-상보적 가닥 둘 모두를 절단하기 위해 감소된 능력을 갖는다. 다른 잔기는 상기 효과를 달성하기 위해 (즉 하나 또는 다른 뉴클레아제 부분을 불활성화시키기 위해) 돌연변이될 수 있다. 비-제한적 예로서, 잔기 D10, G12, G17, E762, H840, N854, N863, H982, H983, A984, D986, 및/또는 A987 (또는 서열번호:1-256 및 795-1346에서 제시된 임의의 단백질의 상응하는 돌연변이)은 변경 (즉, 치환될 수 있다) (참고 Cas9 아미노산 잔기의 보존에 과한 더 많은 정보를 위한 도 3, 도 5, 도 11a, 및 표 1). 또한, 알라닌 치환 이외의 돌연변이가 적당하다. 더 많은 중요한 정보에 대해
표 1. 표 1은 다양한 종으로부터의 Cas9 서열에 존재하는 4 개의 모티프를 열거한다 (또한 하기를 참조한다: 도 3 및 도 5). 본원에서 열거된 아미노산은 S. 파이오제네스로부터의 Cas9이다 (서열번호:8).
일부 경우에서, 키메라 부위 지향적 변형 폴리펩타이드는 아미노산 서열을 포함하고, 이 서열은 도 3에서 묘사된 Cas9/Csn1 아미노산 서열의 아미노산 7-166 또는 731-1003과, 또는 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 상응하는 부분과 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 99% 또는 100% 아미노산 서열 동일성을 갖는다. 일부 경우에서, 키메라 부위 지향적 변형 폴리펩타이드는 4 개의 모티프 (표 4에서 열거되고 도 3a 및 도 5에서 묘사됨)를 포함하고, 아미노산 서열을 갖는 각각은 표 1에서 열거된 각각의 4 개의 모티프(서열번호:260-263)와, 또는 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 상응하는 부분과 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 99% 또는 100% 아미노산 서열 동일성을 갖는다. 일부 경우에서, 키메라 부위 지향적 변형 폴리펩타이드는 아미노산 서열을 포함하고, 이 서열은 도 3에서 묘사된 Cas9/Csn1 아미노산 서열의 아미노산 7-166 또는 731-1003과, 또는 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 상응하는 부분과 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 99% 또는 100% 아미노산 서열 동일성을 갖는다.
일부 구현예에서, 부위 지향적 변형 폴리펩타이드의 활성부는 이종기원 폴리펩타이드를 포함하고, 이것은 DNA-변형 활성 및/또는 전사 인자 활성 및/또는 DNA-연관된 폴리펩타이드-변형 활성을 갖는다. 일부 경우에서, 이종기원 폴리펩타이드는 뉴클레아제 활성을 제공하는 Cas9/Csn1 폴리펩타이드의 부분을 대체한다. 다른 구현예에서, 당해 부위 지향적 변형 폴리펩타이드는 뉴클레아제 활성을 보통 제공하고 (부분은 완전히 활성일 수 있거나 대신에 상응하는 야생형 활성의 100% 미만을 갖도록 변형될 수 있는) Cas9/Csn1 폴리펩타이드의 부분 및 이종기원 폴리펩타이드 둘 모두를 포함한다. 환언하면, 일부 경우에서, 당해 키메라 부위 지향적 변형 폴리펩타이드는 뉴클레아제 활성 및 이종기원 폴리펩타이드를 통상적으로 제공하는 Cas9/Csn1 폴리펩타이드의 부분 둘 모두를 포함하는 융합 폴리펩타이드이다. 다른 사례에서, 당해 키메라 부위 지향적 변형 폴리펩타이드는 Cas9/Csn1 폴리펩타이드의 활성부의 변형된 변이체 (예를 들면, 아미노산 변화, 결실, 삽입) 및 이종기원 폴리펩타이드를 포함하는 융합 폴리펩타이드이다. 또 다른 사례에서, 당해 키메라 부위 지향적 변형 폴리펩타이드는 이종기원 폴리펩타이드 및 자연-발생 또는 변형된 부위 지향적 변형 폴리펩타이드의 RNA-결합부를 포함하는 융합 폴리펩타이드이다.
예를 들면, 키메라 Cas9/Csn1 단백질에서, 자연-발생 (또는 변형된, 예를 들면, 돌연변이, 결실, 삽입) 박테리아 Cas9/Csn1 폴리펩타이드는 이종기원 폴리펩타이드 서열 (즉 Cas9/Csn1 이외의 단백질로부터의 폴리펩타이드 서열 또는 또 하나의 유기체로부터의 폴리펩타이드 서열)에 융합될 수 있다. 이종기원 폴리펩타이드 서열은 키메라 Cas9/Csn1 단백질에 의해 또한 나타날 활성 (예를 들면, 효소 활성) (예를 들면, 메틸전이효소 활성, 아세틸전이효소 활성, 키나아제 활성, 우비퀴틴화 활성, 등)을 나타낼 수 있다. 이종기원 핵산 서열은 키메라 폴리펩타이드를 인코딩하는 키메라 뉴클레오타이드 서열를 산출하기 위해 (예를 들면, 유전공학에 의해) 또 하나의 핵산 서열에 연결될 수 있다. 일부 구현예에서, 키메라 Cas9/Csn1 폴리펩타이드는 준세포 국재화 (예를 들면, 핵을 표적으로 하는 핵 국재화 신호 (NLS); 미토콘드리아를 표적으로 하는 미토콘드리아 국재화 신호; 엽록체를 표적으로 하는 엽록체 국재화 신호; ER 체류 신호; 등)를 제공하는 이종기원 서열로 Cas9/Csn1 폴리펩타이드 (예를 들면, 야생형 Cas9 또는 Cas9 변이체, 예를 들면, 감소된 또는 불활성화된 뉴클레아제 활성을 갖는 Cas9)를 융합하여 산출된다. 일부 구현예에서, 이종기원 서열은 추적 또는 정제의 용이하게 하기 위해 태그를 제공할 수 있다 (예를 들면, 형광 단백질, 예를 들면, 녹색 형광 단백질 (GFP), YFP, RFP, CFP, mCherry, tdTomato, 등; His 태그, 예를 들면, 6XHis 태그; 혈구응집소 (HA) 태그; FLAG 태그; Myc 태그; 등). 일부 구현예에서, 이종기원 서열은 증가된 또는 감소된 안정성을 제공할 수 있다. 일부 구현예에서, 이종기원 서열은 (예를 들면, 관심 또 하나의 단백질, 예를 들면, DNA 또는 히스톤 변형 단백질, 전사 인자 또는 전사 억제인자, 동원 단백질, 등에 결합하도록 키메라 Cas9 폴리펩타이드의 능력을 제공하기 위해) 결합 도메인을 제공할 수 있다.
당해 변이체 Cas9 부위 지향적 폴리펩타이드를 위한 다양한 추가의 적당한 융합 파트너 (또는 그의 단편)의 예는 도 54에서 열거된 것을 비제한적으로 포함한다.
당해 키메라 부위 지향적 변형 폴리펩타이드를 인코딩하는 핵산
당해 개시내용은 뉴클레오타이드 서열을 포함하는 당해 키메라 부위 지향적 변형 폴리펩타이드를 인코딩하는 핵산을 제공한다. 일부 구현예에서, 뉴클레오타이드 서열을 포함하는 당해 키메라 부위 지향적 변형 폴리펩타이드를 인코딩하는 핵산은 발현 벡터, 예를 들면, 재조합 발현 벡터이다.
일부 구현예에서, 당해 방법은 표적 DNA를 접촉시키거나 세포 (또는 세포의 모집단)에 키메라 부위 지향적 변형 폴리펩타이드를 포함하는 하나 이상의 핵산을 도입하는 것을 수반한다. 키메라 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 적당한 핵산은 발현 벡터를 포함하고, 여기서 키메라 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 발현 벡터는 "재조합 발현 벡터"이다.
일부 구현예에서, 재조합 발현 벡터는 바이러스 구조체, 예를 들면, 재조합 아데노-연관된 바이러스 구조체 (참고, 예를 들면, 미국 특허 번호 7,078,387), 재조합 아데노바이러스 구조체, 재조합 렌티바이러스 구조체 등이다.
적당한 발현 벡터는, 비제한적으로, 하기를 포함한다: 바이러스 벡터 (예를 들면 우두 바이러스 기반 바이러스 벡터; 폴리오바이러스; 아데노바이러스 (참고, 예를 들면, Li 등, Invest Opthalmol Vis Sci 35:2543 2549, 1994; Borras 등, Gene Ther 6:515 524, 1999; Li 및 Davidson, PNAS 92:7700 7704, 1995; Sakamoto 등, H Gene Ther 5:1088 1097, 1999; WO 94/12649, WO 93/03769; WO 93/19191; WO 94/28938; WO 95/11984 및 WO 95/00655); 아데노-연관된 바이러스 (참고, 예를 들면, Ali 등, Hum Gene Ther 9:81 86, 1998, Flannery 등, PNAS 94:6916 6921, 1997; Bennett 등, Invest Opthalmol Vis Sci 38:2857 2863, 1997; Jomary 등, Gene Ther 4:683 690, 1997, Rolling 등, Hum Gene Ther 10:641 648, 1999; Ali 등, Hum Mol Genet 5:591 594, 1996; Srivastava in WO 93/09239, Samulski 등, J. Vir. (1989) 63:3822-3828; Mendelson 등, Virol. (1988) 166:154-165; 및 Flotte 등, PNAS (1993) 90:10613-10617); SV40; 단순 포진 바이러스; 인간 면역결핍 바이러스 (참고, 예를 들면, Miyoshi 등, PNAS 94:10319 23, 1997; Takahashi 등, J Virol 73:7812 7816, 1999); 레트로바이러스 벡터 (예를 들면, 쥣과 백혈병 바이러스, 비장 괴사 바이러스, 및 레트로바이러스 예컨대 루(Rous) 육종 바이러스, 하비 육종 바이러스, 조류 백혈증 바이러스, 렌티바이러스, 인간 면역결핍 바이러스, 골수증식성 육종 바이러스, 및 유선 종양 바이러스로부터 유도된 벡터); 등.
수많은 적당한 발현 벡터는 당해분야의 숙련가에게 공지되어 있고, 많은 것들은 상업적으로 이용가능하다. 하기의 벡터는 진핵 숙주 세포에 대해 하기 예로써 제공된다: pXT1, pSG5 (Stratagene), pSVK3, pBPV, pMSG, 및 pSVLSV40 (Pharmacia). 그러나, 임의의 다른 벡터는 숙주세포와 있는 그대로 양립가능한 한 사용될 수 있다.
이용된 숙주/벡터 시스템에 따라, 항시적 및 유도성 프로모터를 포함하는 임의의 수의 적당한 전사 및 번역 조절 요소, 전사 인핸서 요소, 전사 종결자 등은 발현 벡터에서 사용될 수 있다 (참고 예를 들면, Bitter 등 (1987) Methods in Enzymology, 153:516-544).
일부 구현예에서, 키메라 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열은 조절 요소, 예를 들면, 전사 조절 요소, 예컨대 프로모터에 작동가능하게 연결된다. 전사 조절 요소는 진핵 세포, 예를 들면, 포유동물 세포; 또는 원핵 세포 (예를 들면, 박테리아 또는 고세균 세포)에서 기능적일 수 있다. 일부 구현예에서, 키메라 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열은 키메라 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열의 발현을 원핵 및 진핵 세포 둘 모두에서 허용하는 다중 조절 요소에 작동가능하게 연결된다.
적당한 진핵 프로모터 (진핵 세포에서 기능적인 프로모터)의 비-제한적인 예는 사이토메갈로바이러스 (CMV) 전초기, 단순 포진 바이러스 (HSV) 티미딘 키나아제, 초기 및 말기 SV40, 레트로바이러스로부터 긴 말단 반복 (LTR), 및 마우스 메탈로티오네인-I으로부터의 것들을 포함한다. 적절한 벡터 및 프로모터의 선택은 당업자의 수준 내에 있다. 발현 벡터는 번역 개시 및 전사 종결자를 위해 리보솜 결합 부위를 또한 함유할 수 있다. 발현 벡터는 발현을 증폭시키기 위해 적절한 서열을 또한 포함할 수 있다. 발현 벡터는 키메라 부위 지향적 변형 폴리펩타이드에 융합된 뉴클레오타이드 서열 인코딩 단백질 태그 (예를 들면, 6xHis 태그, 혈구응집소 (HA) 태그, 형광 단백질 (예를 들면, 녹색 형광 단백질; 황색 형광 단백질, 등), 등)를 또한 포함할 수 있다.
일부 구현예에서, 키메라 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열은 유도성 프로모터 (예를 들면, 열충격 프로모터, 테트라사이클린-조절된 프로모터, 스테로이드-조절된 프로모터, 금속-조절된 프로모터, 에스트로겐 수용체-조절된 프로모터, 등)에 작동가능하게 연결된다. 일부 구현예에서, 키메라 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열은 공간적으로 제한된 및/또는 일시적으로 제한된 프로모터 (예를 들면, 조직 특이적 프로모터, 세포형 특이적 프로모터, 등) 에 작동가능하게 연결된다. 일부 구현예에서, 키메라 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열은 항시적 프로모터에 작동가능하게 연결된다.
핵산을 숙주세포에 도입하는 방법는 대상체기술에 공지되어 있고, 임의의 공지된 방법은 핵산 (예를 들면, 발현 구조체)을 줄기세포 또는 전구 세포에 도입하기 위해 사용될 수 있다. 적당한 방법은 예를 들면, 바이러스 또는 박테리오파아지 감염, 형질감염, 콘주게이션, 원형질 융합, 리포펙션, 전기천공, 칼슘 포스페이트 침전, 폴리에틸렌이민 (PEI)-매개된 형질감염, DEAE-덱스트란 매개된 형질감염, 리포좀-매개된 형질감염, 입자총 기술, 칼슘 포스페이트 침전, 직접적인 현미 주입, 나노입자-매개된 핵산 전달을 포함한다 (참고, 예를 들면, Panyam 등, Adv Drug Deliv Rev. 2012 Sep 13. pii: S0169-409X(12)00283-9. doi: 10.1016/j.addr.2012.09.023), 등.
방법
당해 개시내용은 표적 DNA 및/또는 표적 DNA-연관된 폴리펩타이드를 변형시키는 방법을 제공한다. 일반적으로, 당해 방법은 표적 DNA를 복합물 ("표적 복합물 ")과 접촉시키는 것을 수반하고, which 이 복합물은 DNA-표적화 RNA 및 부위 지향적 변형 폴리펩타이드를 포함한다.
상기에서 논의된 바와 같이, 당해 DNA-표적화 RNA 및 당해 부위 지향적 변형 폴리펩타이는 복합물을 형성한다. DNA-표적화 RNA는 표적 DNA의 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하여 복합물에 대한 표적 특이성을 제공한다. 복합물의 부위 지향적 변형 폴리펩타이드는 부위 특이적인 활성을 제공한다. 일부 구현예에서, 당해 복합물은, 예를 들면, DNA 절단, DNA 메틸화, DNA 손상, DNA 치유 등으로 인도되는 표적 DNA를 변형시킨다. 다른 구현예에서, 당해 복합물은, 예를 들면, 히스톤 메틸화, 히스톤 아세틸화, 히스톤 우비퀴틴화, 등으로 인도되는 표적 DNA와 연관된 표적 폴리펩타이드 (예를 들면, 히스톤, DNA-결합 단백질, 등)를 변형시킨다. 표적 DNA는, 예를 들면, 시험관내의 기존의 DNA, 시험관 내의 세포 중 염색체 DNA, 생체 내의 세포 중 염색체 DNA 등일 수 있다.
일부 경우에서, 부위 지향적 변형 폴리펩타이드는 DNA-표적화 RNA와 표적 DNA 사이의 상보성의 영역에 의해 규정된 표적 DNA 서열에서 표적 DNA를 절단하는 뉴클레아제 활성을 나타낸다. 일부 경우에서, 부위 지향적 변형 폴리펩타이드는 Cas9 또는 Cas9 관련된 폴리펩타이드일 때, 표적 DNA의 부위 특이적인 절단은 하기의 둘 모두에서 결정된 위치에서 일어난다: (i) DNA-표적화 RNA와 표적 DNA 사이의 염기 짝짓기 상보성; 및 (ii) 표적 DNA 중 짧은 모티프 [프로토스페이서 인접한 모티프 (PAM)라 칭함]. 일부 구현예에서 (예를 들면, S. 파이오제네스로부터의 Cas9, 또는 밀접하게 관련된 Cas9가 사용될 때 (참고 서열번호:1-256 및 795-1346)), 비-상보적 가닥의 PAM 서열은 5'-XGG-3'이고, 여기서 X는 임의의 DNA 뉴클레오타이드이고 X는 비-표적화 DNA의 상보적 가닥의 표적 서열의 3' 바로 옆에 있다 (참고 도 10). 이와 같이, 상보적 가닥의 PAM 서열은 5'-CCY-3'이고, 여기서 Y는 임의의 DNA 뉴클레오타이드이고 Y는 표적 DNA의 상보적 가닥의 표적 서열의 5' 바로 옆에 있다 (참고 도 10, 여기서 비-상보적 가닥의 PAM은 5'-GGG-3'이고 상보적 가닥의 PAM은 5'-CCC-3'이다). 그와 같은 일부 구현예에서, X 및 Y는 상보적일 수 있고 X-Y 염기쌍은 임의의 염기쌍일 수 있다 (예를 들면, X=C 및 Y=G; X=G 및 Y=C; X=A 및 Y=T, X=T 및 Y=A).
일부 경우에서, 상이한 Cas9 단백질 (즉, 다양한 종으로부터의 Cas9 단백질)은 상이한 Cas9 단백질의 다양한 효소 특성을 자본화하기 위해 다양한 제공된 방법에서 사용하는데 유리할 수 있다 (예를 들면, 상이한 PAM 서열 선호에 대해; 증가된 또는 감소된 효소 활성에 대해; 세포 독성의 증가된 또는 감소된 수준에 대해; NHEJ, 상동성-유도된 치유, 단일 가닥 절단, 이중 가닥 절단, 등 사이에 균형을 변화시키기 위해). 다양한 종으로부터의 Cas9 단백질 (참고 서열번호:1-256 및 795-1346)은 표적 DNA에서 상이한 PAM 서열을 필요로 할 수 있다. 따라서, 선택의 특정한 Cas9 단백질에 대해, PAM 서열 요건은 상기 기재된 5'-XGG-3' 서열과 상이할 수 있다.
다양한 종으로부터 이동상동성인 많은 Cas9가 본원에서 확인되었고 단백질은 단지 몇 개의 동일한 아미노산을 공유한다. 모든 확인된 Cas9 오쏘로그는 중심 HNH 엔도뉴클레아제 도메인을 갖는 동일한 도메인 구조를 가지며 RuvC/RNAeH 도메인을 분리한다 (참고 도 3A, 3B, 도 5, 및 표 1). Cas9 단백질은 보존된 구조를 갖는 4 개의 주요 모티프를 공유한다. 모티프 1, 2, 및 4는 RuvC 예컨대 모티프이고, 한편 모티프 3은 HNH-모티프이다. 일부 경우에서, 적당한 부위 지향적 변형 폴리펩타이드는 아미노산 서열을 포함하고, 이 서열은 4 개의 모티프를 가지며, 각각의 모티프 1-4는 도 3a에서 묘사된 Cas9/Csn1 아미노산 서열 (서열번호:260-263, 각각은 표 1에서 묘사됨)의 모티프 1-4에 대해, 또는 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 상응하는 부분 (참고 분기형 Cas9 서열로부터의 모티프 1-4의 정렬에 대한 도 5)에 대해 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 99% 또는 100% 아미노산 서열 동일성을 갖는다. 일부 경우에서, 적당한 부위 지향적 변형 폴리펩타이드는 아미노산 서열을 포함하고, 이 서열은 도 3에서 묘사된 Cas9/Csn1 아미노산 서열의 아미노산 7-166 또는 731-1003과, 또는 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 상응하는 부분과 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 99% 또는 100% 아미노산 서열 동일성을 갖는다. 상기에서 규정된 바와 같은 임의의 Cas9 단백질은 부위 지향적 변형 폴리펩타이드로서 또는 본 발명의 키메라 부위 지향적 변형 폴리펩타이드의 부분으로서 사용될 수 있다.
뉴클레아제 활성은 이중 가닥 절단을 생산하기 위해 표적 DNA를 절단한다. 그 다음 이들 절단은 하기의 2 개의 방식 중 하나에서 세포에 의해 치유된다: 비-상동 말단 연결, 및 상동성-유도된 치유 (도 2). 비-상동 말단 연결 (NHEJ)에서, 이중-가닥 절단은 서로에 대한 절단 말단의 직접적인 결찰에 의해 치유된다. 이와 같이, 신규 핵산 물질은 그 부위에 삽입되지 않지만, 일부 핵산 물질은 잃을 수 있고, 그 결과 결실이 생긴다. 상동성-유도된 치유에서, 절단된 표적 DNA 서열에 대한 상동성을 갖는 공여체 폴리뉴클레오타이드는 절단된 표적 DNA 서열의 치유에 대해 템플레이트로서 사용되고, 그 결과, 공여체 폴리뉴클레오타이드로부터 표적 DNA로 유전적 정보가 전달된다. 이와 같이, 신규 핵산 물질은 그 부위로 삽입/복사될 수 있다. 일부 경우에서, 표적 DNA는 당해 공여체 폴리뉴클레오타이드와 접촉된다. 일부 경우에서, 당해 공여체 폴리뉴클레오타이드는 당해 세포로 도입된다. NHEJ로 인한 표적 DNA의 변형 및/또는 상동성-유도된 치유는, 예를 들면, 유전자 수정, 유전자 대체, 유전자 태깅, 이식유전자 삽입, 뉴클레오타이드 결실, 유전자 파괴, 유전자 돌연변이 등으로 이어진다.
따라서, 부위 지향적 변형 폴리펩타이드에 의한 DNA의 절단은 표적 DNA 서열을 절단하고 외인성으로 제공된 공여체 폴리뉴클레오타이드의 부재에서 세포가 서열을 치유하도록 하여 표적 DNA 서열로부터 핵산 물질을 결실시키기 위해 (예를 들면, 세포를 감염에 대해 민감하도록 하는 유전자 (예를 들면 T 세포를 HIV 감염에 대해 민감하도록 하는 CCR5 또는 CXCR4 유전자)를 파괴하고, 뉴런에서 질환-야기 트리뉴클레오타이드 반복 서열을 제거하고, 연구에서 질환 모델로서 유전자 녹아웃 및 돌연변이를 창출하기 위해, 등) 사용될 수 있다. 따라서, 당해 방법은 유전자 (이로써 전사 또는 변경된 전사가 완전히 없어진다)을 녹 아웃(knock out)하기 위해서나 표적 DNA에서 선택된 장소로 유전적 물질을 녹 인(knock in)하기 위해 사용될 수 있다.
대안적으로, DNA-표적화 RNA 및 부위 지향적 변형 폴리펩타이드가 표적 DNA 서열에 대한 상동성을 갖는 분절을 적어도 포함하는 공여체 폴리뉴클레오타이드 서열과 함께 세포에 공투여되면, 당해 방법은, 핵산 물질을 표적 DNA 서열에 부가, 즉 삽입 또는 대체하기 위해 (예를 들면 단백질, siRNA, miRNA, 등을 인코딩하는 핵산을 "녹 인(knock in)"하기 위해), 태그 (예를 들면, 6xHis, 형광 단백질 (예를 들면, 녹색 형광 단백질; 황색 형광 단백질, 등)를 부가하기 위해, 혈구응집소 (HA), FLAG, 등), 조절 서열을 유전자 (예를 들면 프로모터, 폴리아데닐화 신호, 내부 리보솜 진입 서열 (IRES), 2A 펩타이드, 시작 코돈, 정지 코돈, 스플라이스 신호, 국재화 신호, 등)에 부가하기 위해, 핵산 서열을 변형시키기 위해 (예를 들면, 돌연변이를 도입하는 등을 위해) 사용될 수 있다. 이와 같이, DNA-표적화 RNA 및 부위 지향적 변형 폴리펩타이드를 포함하는 복합물은 임의의 시험관내 또는 생체내 적용에서 유용하고 여기서 예를 들면, 질환을 치료하기 위한 유전자 요법에서 사용되는 바와 같이 또는 항바이러스, 항병원체, 또는 항암 치료, 농업에서 유전적으로 변형된 유기체의 생산, 치료, 진단, 또는 연구 목적을 위한 세포에 의한 단백질의 대규모 생산, iPS 세포의 유도, 생물학적 연구, 결실 또는 대체에 대한 병원체의 유전자의 표적화, 등으로서 부위 특이적인, 즉 "표적화된" 방식, 예를 들면 유전자 넉아웃, 유전자 녹인(knock-in), 유전자 편집, 유전자 태깅, 등에서 DNA를 변형시키는 것이 바람직하다.
일부 구현예에서, 부위 지향적 변형 폴리펩타이드는 변형된 형태의 Cas9/Csn1 단백질을 포함한다. 일부 예에서, 변형된 형태의 Cas9/Csn1 단백질은 Cas9/Csn1 단백질의 자연-발생 뉴클레아제 활성을 감소시키는 아미노산 변화 (예를 들면, 결실, 삽입, 또는 치환)를 포함한다. 예를 들면, 일부 예에서, 변형된 형태의 Cas9/Csn1 단백질은 상응하는 야생형 Cas9/Csn1 폴리펩타이드의 뉴클레아제 활성의 50% 미만, 40% 미만, 30% 미만, 20% 미만, 10% 미만, 5% 미만, 또는 1% 미만을 갖는다. 일부 경우에서, 변형된 형태의 Cas9/Csn1 폴리펩타이드는 실질적인 뉴클레아제 활성을 갖지 않는다. 당해 부위 지향적 변형 폴리펩타이드가 실질적인 뉴클레아제 활성을 갖지 않는 변형된 형태의 Cas9/Csn1 폴리펩타이드일 때, "dCas9"라 칭할 수 있다.
일부 구현예에서, 변형된 형태의 Cas9/Csn1 폴리펩타이드는 D10A (서열번호:8의 아미노산 위치 10에서 아스파르테이트 내지 알라닌) 돌연변이 (또는 서열번호:1-256 및 795-1346에서 제시된 임의의 단백질의 상응하는 돌연변이)이고, 이것은 표적 DNA의 상보적 가닥을 절단할 수 있지만 비-표적화 DNA의 상보적 가닥을 절단하기 위한 감소된 능력을 갖는다 (따라서 DSB 대신에 단일 가닥 절단 (SSB)이 생긴다; 참고 도 11). 일부 구현예에서, 변형된 형태의 Cas9/Csn1 폴리펩타이드는 H840A (서열번호:8의 아미노산 위치 840에서 히스티딘 내지 알라닌) 돌연변이 (또는 서열번호:1-256 및 795-1346에서 제시된 임의의 단백질의 상응하는 돌연변이)이고, 이것은 비-표적화 DNA의 상보적 가닥을 절단할 수 있지만 표적 DNA의 상보적 가닥을 절단하기 위한 감소된 능력을 갖는다 (따라서 DSB 대신에 단일 가닥 절단 (SSB)이 생긴다; 참고 도 11). Cas9의 D10A 또는 H840A 변이체 (또는 서열번호:1-256 및 795-1346에서 제시된 임의의 단백질에서 상응하는 돌연변이)의 사용이 기대된 생물학적 결과를 변경시킬 수 있는 것은, 비-상동 말단 연결 (NHEJ)이 DSB가 SSB와는 반대로 존재할 때 훨씬 더 일어날 것 같기 때문이다. 따라서, 일부 경우에서 DSB의 가능성을 감소시키고 (따라서 NHEJ의 가능성을 감소)시키기를 원할 때, Cas9의 D10A 또는 H840A 변이체가 사용될 수 있다. 다른 잔기는 동일한 효과를 달성하기 위해 (즉 하나 또는 다른 뉴클레아제 부분을 불활성화)시키기 위해 돌연변이될 수 있다. 비-제한적 예로서, 잔기 D10, G12, G17, E762, H840, N854, N863, H982, H983, A984, D986, 및/또는 A987 (또는 서열번호:1-256 및 795-1346에서 제시된 임의의 단백질의 상응하는 돌연변이)은 변경 (즉, 치환)될 수 있다 (참고 Cas9 아미노산 잔기의 보존에 관한 더 많은 정보에 대한 도 3, 도 5, 도 11a, 및 표 1). 또한, 알라닌 치환 이외의 돌연변이가 적당하다. 일부 구현예에서 부위 지향적 폴리펩타이드 (예를 들면, 부위-지향된 변형 폴리펩타이드)가 감소된 촉매 활성을 가질 때 (예를 들면, Cas9 단백질이 D10, G12, G17, E762, H840, N854, N863, H982, H983, A984, D986, 및/또는 A987 돌연변이, 예를 들면, D10A, G12A, G17A, E762A, H840A, N854A, N863A, H982A, H983A, A984A, 및/또는 D986A를 가질 때), 폴리펩타이드는, DNA-표적화 RNA와 상호작용하는 능력을 보유하는 한 부위 특이적인 방식에서 표적 DNA을 알지 못할 수 있다 (그 이유는 DNA-표적화 RNA에 의해 표적 DNA 서열로 또한 유도되기 때문이다).
일부 구현예에서, 변형된 형태의 Cas9/Csn1 폴리펩타이드는 D10A 및 H840A 돌연변이 (또는 서열번호:1-256 및 795-1346에서 제시된 임의의 단백질의 상응하는 돌연변이) 둘 모두 제공하고, 이로써 폴리펩타이드는 표적화 DNA의 상보적 및 비-상보적 가닥 둘 모두를 절단하기 위해 감소된 능력을 갖는다 (즉, 변이체는 실질적인 뉴클레아제 활성을 가질 수 없다). 다른 잔기는 동일한 효과를 제공하기 위해 (즉 하나 또는 다른 뉴클레아제 부분을 불활성화시키기 위해) 돌연변이될 수 있다. 비-제한적 예로서, 잔기 D10, G12, G17, E762, H840, N854, N863, H982, H983, A984, D986, 및/또는 A987 (또는 서열번호:1-256 및 795-1346에서 제시된 임의의 단백질의 상응하는 돌연변이)가 변경 (즉, 치환될 수 있다) (참고 Cas9 아미노산 잔기의 보존에 관한 더 많은 정보에 대한 도 3, 도 5, 도 11a, 및 표 1). 또한, 알라닌 치환 이외의 돌연변이가 적당하다.
일부 구현예에서, 부위 지향적 변형 폴리펩타이드는 이종기원 서열 (예를 들면, 융합)을 포함한다. 일부 구현예에서, 이종기원 서열은 부위 지향적 변형 폴리펩타이드의 준세포 국재화 (예를 들면, 핵을 표적으로 하는 핵 국재화 신호 (NLS); 미토콘드리아를 표적으로 하는 미토콘드리아 국재화 신호; 엽록체를 표적으로 하는 엽록체 국재화 신호; ER 체류 신호; 등)를 제공할 수 있다. 일부 구현예에서, 이종기원 서열은 추적 또는 정제의 용이하게 하기 위해 태그를 제공할 수 있다 (예를 들면, 형광 단백질, 예를 들면, 녹색 형광 단백질 (GFP), YFP, RFP, CFP, mCherry, tdTomato, 등; His 태그, 예를 들면, 6XHis 태그; 혈구응집소 (HA) 태그; FLAG 태그; Myc 태그; 등). 일부 구현예에서, 이종기원 서열은 증가된 또는 감소된 안정성을 제공할 수 있다.
일부 구현예에서, 당해 부위 지향적 변형 폴리펩타이드는 코돈-최적화될 수 있다. 이러한 유형의 최적화는 대상체기술에서 공지되어 있고 동일한 단백질을 인코딩하면서 의도된 숙주 유기체 또는 세포의 코돈 신호를 모방하기 위해 이물질-유도된 DNA의 돌연변이를 수반한다. 따라서, 코돈은 변화되지만, 인코딩된 단백질은 변화되지 않은 채로 있다. 예를 들면, 의도된 표적 세포가 인간 세포였다면, 인간 코돈-최적화된 Cas9 (또는 변이체, 예를 들면, 효소적으로 불활성 변이체)는 적당한 부위 지향적 변형 폴리펩타이드이다 (참고 예로써 서열번호:256). 임의의 적당한 부위 지향적 변형 폴리펩타이드 (예를 들면, 임의의 Cas9 예컨대 서열번호:1-256 및 795-1346에서 제시된 임의의 서열)은 코돈 최적화될 수 있다. 또 하나의 비-제한적인 예로서, 의도된 숙주세포가 마우스 세포였다면, 마우스 코돈-최적화된 Cas9 (또는 변이체, 예를 들면, 효소적으로 불활성 변이체)는 적당한 부위 지향적 변형 폴리펩타이드이다. 코돈 최적화가 필요한 것은 아니지만, 허용가능하고 어떤 경우에 바람직할 수 있다.
일부 구현예에서, 당해 DNA-표적화 RNA 및 당해 부위 지향적 변형 폴리펩타이드는 박테리아 세포에서 유전자 발현을 차단하기 위한 유도성 시스템으로서 사용된다. 일부 경우에서, 적절한 DNA-표적화 RNA 및/또는 적절한 부위 지향적 폴리펩타이드를 인코딩하는 핵산은 표적 세포의 염색체에 편입되고 유도성 프로모터의 제어 하에 있다. DNA-표적화 RNA 및/또는 부위 지향적 폴리펩타이드가 유도될 때, 표적 DNA는, DNA-표적화 RNA 및 부위 지향적 변형 폴리펩타이드 둘 모두가 존재하고 복합물을 형성할 때 관심 위치 (예를 들면, 별개의 플라스미드 상의 표적 유전자)에서 절단 (또는 변형)된다. 이와 같이, 일부 경우에서, 박테리아 발현 균주는 (예를 들면, 유도성 프로모터의 제어 하에서) 플라스미드 상의 적절한 DNA-표적화 RNA 및/또는 박테리아 게놈에서 적절한 부위 지향적 변형 폴리펩타이드를 인코딩하는 핵산 서열을 포함하도록 조작되고, 이는 (균주에 도입된 별개의 플라스미드로부터 발현된) 임의의 표적화된 유전자의 발현이 DNA-표적화 RNA 및 부위 지향적 폴리펩타이드의 발현을 유도하여 조절되는 실험을 허용한다.
일부 경우에서, 부위 지향적 변형 폴리펩타이드는 이중 가닥 절단을 도입하는 것 외의 방식으로 표적 DNA를 변형시키는 효소 활성을 갖는다. 표적 DNA (예를 들면, 부위 지향적 변형 폴리펩타이드에 대한 효소 활성 이종기원 폴리펩타이드를 융합하여, 그렇게 함으로써 키메라 부위 지향적 변형 폴리펩타이드를 산출한다)를 변형시키도록 사용될 수 있는 관심 효소 활성은, 비제한적으로 하기를 포함한다: 메틸전이효소 활성, 탈메틸효소 활성, DNA 치유 활성, DNA 손상 활성, 탈아미노화 활성, 디스무타제 활성, 알킬화 활성, 탈퓨린화 활성, 산화 활성, 피리미딘 이량체 형성 활성, 인테그라제 활성, 전위효소 활성, 재조합효소 활성, 폴리머라제 활성, 리가제 활성, 헬리카제 활성, 광분해효소 활성 또는 글리코실라제 활성). 메틸화 및 데메틸화는 우성유전 유전자 조절의 중요한 방식으로 대상체기술에서 인식되어 있지만, DNA 손상 및 치유 활성은 환경적인 스트레스에 반응하여 세포 생존 및 적절한 게놈 유지에 대해 필수적이다.
이와 같이, 본 방법은 표적 DNA의 후성유전적 변형에서의 용도를 발견하고 원하는 상보적 핵산 서열을 DNA-표적화 RNA의 DNA-표적화 분절로 유전적으로 조작하여 표적 DNA의 임의의 위치에서 표적 DNA의 후성유전적 변형을 제어하도록 이용될 수 있다. 본원의 방법은 또한 표적 DNA 내의 임의의 원하는 위치에서 DNA의 의도적인 및 조절된 손상세어의 용도를 발견한다. 본원의 방법은 또한 표적 DNA 내의 임의의 원하는 위치에서 DNA의 서열-특이적 및 조절된 치유에서의 용도를 발견한다. DNA-변형 효소 활성을 표적 DNA의 특이적 위치로 표적화하는 방법은 연구 및 임상 적용 둘 모두에서의 용도를 발견한다.
일부 경우에서, 부위 지향적 변형 폴리펩타이드는 (예를 들면, 키메라 부위 지향적 변형 폴리펩타이드, 등의 경우에) 표적 DNA의 전사를 조절하는 활성을 갖는다. 일부 경우에서, 전사를 증가 또는 감소시키는 능력을 나타내는 이종기원 폴리펩타이드 (예를 들면, 전사 활성제 또는 전사 억제인자 폴리펩타이드)를 포함하는 키메라 부위 지향적 변형 폴리펩타이드는 DNA-표적화 RNA의 DNA-표적화 분절에 의해 유도된 표적 DNA의 특이적 위치에서 표적 DNA의 전사를 증가 또는 감소시키기 위해 사용된다. 키메라 부위 지향적 변형 폴리펩타이드에 전사 조절 활성을 제공하기 위한 공급원 폴리펩타이드의 예는, 비제한적으로 광-유도성 전사 조절물질, 소분자/약물-반응성 전사 조절물질, 전사 인자, 전사 억제인자, 등을 포함한다. 일부 경우에서, 당해 방법은 표적화된 코딩-RNA (단백질-인코딩 유전자) 및/또는 표적화된 비-코딩 RNA (예를 들면, tRNA, rRNA, snoRNA, siRNA, miRNA, long ncRNA, 등)의 발현을 제어하기 위해 사용된다.
일부 경우에서, 부위 지향적 변형 폴리펩타이드는 DNA와 연관된 폴리펩타이드를 변형시키는 효소 활성을 갖는다 (예를 들면 히스톤). 일부 구현예에서, 효소 활성은 하기이다: 메틸전이효소 활성, 탈메틸효소 활성, 아세틸전이효소 활성, 탈아세틸화효소 활성, 키나아제 활성, 포스파타제 활성, 우비퀴틴 리가제 활성 (즉, 우비퀴틴화 활성), 탈우비퀴틴화 활성, 아데닐화 활성, 탈아데닐화 활성, 스모화 활성, 탈스모화 활성, 리보실화 활성, 탈리보실화 활성, 미리스토일화 활성, 탈미리스토일화 활성 당화 활성 (예를 들면, O-GlcNAc 전이효소로부터) 또는 탈당화 활성. 본원에서 열거된 효소 활성은 단백질에 대한 공유 변형을 촉진한다. 그와 같은 변형은 표적 단백질의 안정성 또는 활성을 변경하기 위한 대상체기술에 공지되어 있다 (예를 들면, 키나아제 활성으로 인한 인산화는 표적 단백질에 따라 단백질 활성을 자극 또는 사일런싱할 수 있다). 단백질로서 특정한 관심 중에서 표적은 히스톤이다. 히스톤 단백질은 DNA를 결합하고 뉴클레오솜으로서 공지된 복합체를 형성하기 위해 대상체기술에서 공지되어 있다. 히스톤은 주위의 DNA에서 구조적 변화를 유도하기 위해 (예를 들면, 메틸화, 아세틸화, 유비퀴틴화, 인산화에 의해) 변형될 수 있고, 따라서 상호작용 인자 예컨대 전사 인자, 폴리머라제 등에 대한 잠재적으로 큰 부분의 DNA의 접근성을 조절한다. 단일 히스톤은 많은 상이한 방식으로 그리고 많은 상이한 조합에서 변형될 수 있다 (예를 들면, 히스톤 3의 라이신 27의 트리메틸화, H3K27은, 재프레싱된 전사의 DNA 영역과 연관되지만, 히스톤 3의 라이신 4의 트리메틸화, H3K4는 활성 전사의 DNA 영역과 연관된다). 따라서, 히스톤-변형 활성을 갖는 부위 지향적 변형 폴리펩타이드는 DNA 구조의 부위 특이적 조절에서의 용도를 발견하고 표적 DNA의 선택된 영역에서 히스톤 변형 패턴을 변경시키시 위해 사용될 수 있다. 그와 같은 방법은 연구 및 임상 적용 둘 모두에서의 용도를 발견한다.
일부 구현예에서, 다중 DNA-표적화 RNA는 동일한 표적 DNA 또는 상이한 표적 DNA 상의 상이한 위치를 동시에 변형시키기 위해 동시에 사용된다. 일부 구현예에서, 2 이상의 DNA-표적화 RNA는 동일한 유전자 또는 전사체 또는 장소를 표적으로 한다. 일부 구현예에서, 2 이상의 DNA-표적화 RNA는 상이한 관련없는 유전자좌위를 표적으로 한다. 일부 구현예에서, 2 이상의 DNA-표적화 RNA는 상이하지만, 관련된 유전자좌위를 표적으로 한다.
일부 경우에서, 부위 지향적 변형 폴리펩타이드는 단백질로서 직접적으로 제공된다. 하나의 비제한적인 예로서, 진균 (예를 들면, 효모)는 스페로플라스트 형질전환을 사용하여 외인성 단백질 및/또는 핵산으로 형질전환될 수 있다 (참고 Kawai 등, Bioeng Bugs. 2010 Nov-Dec;1(6):395-403 : "Transformation of Saccharomyces cerevisiae and other fungi: methods and possible underlying mechanism"; 및 Tanka 등, Nature. 2004 Mar 18;428(6980):323-8: "Conformational variations in an infectious protein determine prion strain differences"; 이 둘 모두는 그 전체가 참조로 본원에 편입되어 있다). 따라서, 부위 지향적 변형 폴리펩타이드 (예를 들면, Cas9)는 (DNA-표적화 RNA를 인코딩하는 핵산의 유무에 따라 및 공여체 폴리뉴클레오타이드의 유무에 따라) 스페로플라스트에 편입될 수 있고 스페로플라스트는 내용물을 효모 세포에 도입하기 위해 사용될 수 있다. 부위 지향적 변형 폴리펩타이드는 임의의 편리한 방법에 의해 세포에 도입 (세포에 제공)될 수 있다; 그와 같은 방법은 당해분야의 숙련가에게 공지된다. 또 하나의 비-제한적인 예로서, 부위 지향적 변형 폴리펩타이드는 (예를 들면, DNA-표적화 RNA를 인코딩하는 핵산의 유무에 따라 및 공여체 폴리뉴클레오타이드의 유무에 따라) 세포에, 예를 들면, 제브라피쉬 배아의 세포, 수정된 마우스 난모세포의 전핵 등에 직접적으로 주입될 수 있다.
관심 표적 세포
상기 적용의 일부에서, 당해 방법은 (예를 들면, 개체에 재도입될 수 있는 유전적으로 변형된 세포를 생산하기 위해) 생체내 및/또는 생체외 및/또는 시험관내의 유사분열 또는 후-유사분열 세포에서 DNA 절단, DNA 변형, 및/또는 전사 조절을 도입하기 위해 시용될 수 있다. DNA-표적화 RNA는 표적 DNA를 혼성화하여 특이성을 제공하기 때문에, 개시된 방법 중 관심 유사분열 및/또는 후-유사분열 세포는 하기를 포함할 수 있다: 임의의 유기체로부터의 세포 (예를 들면 박테리아 세포, 고세균 세포, 단일-세포 진핵 유기체의 세포, 식물 세포, 조류 세포, 예를 들면, 보트라이오코쿠스 브라우니이 , 클라마이도모나스 레인하르드티이 , 난노클로롭시스 가디타나, 클로렐라 파이레노이도사, 사르가섬 파텐스 C. 아가르드, 등, 진균 세포 (예를 들면, 효모 세포), 동물 세포, 무척추동물 동물 (예를 들면 초파리, 자포동물, 극피동물, 선충, 등)로부터의 세포, 척추동물 동물 (예를 들면, 물고기, 양서류, 파충류, 새, 포유동물)로부터의 세포, 포유동물로부터의 세포, 설치류로부터의 세포, 인간으로부터의 세포, 등).
임의의 유형의 세포는 관심있는 하기일 수 있다: (예를 들면 줄기세포, 예를 들면 배아 줄기 (ES) 세포, 유도 만능 줄기 (iPS) 세포, 생식세포; 체세포, 예를 들면 섬유아세포, 조혈 세포, 뉴런, 근육 세포, 골 세포, 간세포, 췌장 세포; 임의의 단계, 예를 들면, 1-세포, 2-세포, 4-세포, 8-세포, 등 단계에서의 배아의 시험관내 또는 생체내 배아 세포, 제브라피쉬 배아; 등). 세포는 확립된 세포주로부터 유래될 수 있거나 일차 세포일 수 있고, 여기서 "일차 세포", "일차 세포주", 및 "일차 배양물"은 본원에서 상호교환적으로 사용되고 대상체로부터 유도되었고 배양물의 제한된 수의 계대, 즉 분리 동안에 시험관 내에서의 성장이 허용된 세포 및 세포 배양물을 의미한다. 예를 들면, 일차 배양물은 0 회, 1 회,2 회, 4 회, 5 회, 10 회, 또는 15 회 계대되지만, 위기 단계를 지날 충분한 횟수는 아닌 배양물이다. 전형적으로, 본 발명이 일차 세포주는 시험관 내에서 10 개 미만의 계대 동안에 유지된다. 표적 세포는 많은 구현예에서 단세포 유기체이거나, 배양물에서 성장된다.
세포가 일차 세포이면, 임의의 편리한 방법에 의해 개체로부터 수확될 수 있다. 예를 들면, 백혈구는 분리반출법, 백혈구분리반출법, 밀도 구배 분리, 등에 의해 편리하게 수학될 수 있고, 한편, 조직 예컨대 피부, 근육, 골수, 비장, 간, 췌장, 폐, 창자, 위, 등으로부터의 세포는 생검에 의해 가장 편리하게 수확될 수 있다. 적절한 용액은 수확된 세포의 분산 또는 서스펜션을 위해 사용될 수 있다. 그와 같은 용액은 일반적으로 평형 염액, 예를 들면 정상 식염수, 포스페이트-완충된 염수 (PBS), 행크의 평형 염액, 등일 것이고, 이들은 저농도, 일반적으로 5-25 mM에서의 허용가능한 완충제와 함께 소태아 혈청 또는 다른 자연 발생 인자로 편리하게 보강된다. 편리한 완충제는 HEPES, 포스페이트 완충제, 락테이트 완충제 등을 포함한다. 세포는 즉시 사용될 수 있거나, 장기간 동안 저장되고, 냉동되고, 녹고 재사용될 수 있다. 그와 같은 경우에, 세포는 보통, 그와 같은 냉동 온도에서 세포를 보존하기 위해 대상체기술에서 통상적으로 사용된 바와 같이 10% DMSO, 50% 혈청, 40% 완충된 배지, 또는 일부 다른 그와 같은 용액에서 냉동될 것이고, 냉동된 배양 세포를 용해하기 위해 대상체기술에서 통상적으로 공지된 바와 같이 녹는다.
당해 DNA - 표적화 RNA 및/또는 당해 부위 지향적 변형 폴리펩타이드를 인코딩하는 핵산
일부 구현예에서, 당해 방법은 표적 DNA를 접촉시키거나 세포 (또는 세포의 모집단)에 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 하나 이상의 핵산을 도입하는 것을 수반한다. DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 적당한 핵산은 발현 벡터를 포함하고, 여기서 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 발현 벡터는 "재조합 발현 벡터"이다.
일부 구현예에서, 재조합 발현 벡터는 바이러스 구조체, 예를 들면, 재조합 아데노-연관된 바이러스 구조체 (참고, 예를 들면, 미국 특허 번호 7,078,387), 재조합 아데노바이러스 구조체, 재조합 렌티바이러스 구조체 등이다.
적당한 발현 벡터는, 비제한적으로, 하기를 포함한다: 바이러스 벡터 (예를 들면 우두 바이러스 기반 바이러스 벡터; 폴리오바이러스; 아데노바이러스 (참고, 예를 들면, Li 등, Invest Opthalmol Vis Sci 35:2543 2549, 1994; Borras 등, Gene Ther 6:515 524, 1999; Li 및 Davidson, PNAS 92:7700 7704, 1995; Sakamoto 등, H Gene Ther 5:1088 1097, 1999; WO 94/12649, WO 93/03769; WO 93/19191; WO 94/28938; WO 95/11984 및 WO 95/00655); 아데노-연관된 바이러스 (참고, 예를 들면, Ali 등, Hum Gene Ther 9:81 86, 1998, Flannery 등, PNAS 94:6916 6921, 1997; Bennett 등, Invest Opthalmol Vis Sci 38:2857 2863, 1997; Jomary 등, Gene Ther 4:683 690, 1997, Rolling 등, Hum Gene Ther 10:641 648, 1999; Ali 등, Hum Mol Genet 5:591 594, 1996; Srivastava in WO 93/09239, Samulski 등, J. Vir. (1989) 63:3822-3828; Mendelson 등, Virol. (1988) 166:154-165; 및 Flotte 등, PNAS (1993) 90:10613-10617); SV40; 단순 포진 바이러스; 인간 면역결핍 바이러스 (참고, 예를 들면, Miyoshi 등, PNAS 94:10319 23, 1997; Takahashi 등, J Virol 73:7812 7816, 1999); 레트로바이러스 벡터 (예를 들면, 쥣과 백혈병 바이러스, 비장 괴사 바이러스, 및 레트로바이러스 예컨대 루(Rous) 육종 바이러스, 하비 육종 바이러스, 조류 백혈증 바이러스, 렌티바이러스, 인간 면역결핍 바이러스, 골수증식성 육종 바이러스, 및 유선 종양 바이러스로부터 유도된 벡터); 등.
수많은 적당한 발현 벡터는 당해분야의 숙련가에게 공지되어 있고, 많은 것들은 상업적으로 이용가능하다. 하기의 벡터는 진핵 숙주 세포에 대해 하기 예로써 제공된다: pXT1, pSG5 (Stratagene), pSVK3, pBPV, pMSG, 및 pSVLSV40 (Pharmacia). 그러나, 임의의 다른 벡터는 숙주세포와 있는 그대로 양립가능한 한 사용될 수 있다.
일부 구현예에서, DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열은 조절 요소, 예를 들면, 전사 조절 요소, 예컨대 프로모터에 작동가능하게 연결된다. 전사 조절 요소는 진핵 세포, 예를 들면, 포유동물 세포, 또는 원핵 세포 (예를 들면, 박테리아 또는 고세균 세포)에서 기능성일 수 있다. 일부 구현예에서, DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열은 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열의 발현을 원핵 및 진핵 세포 둘 모두에서 허용하는 다중 조절 요소에 작동가능하게 연결된다.
이용된 숙주/벡터 시스템에 따라, 항시적 및 유도성 프로모터를 포함하는 임의의 수의 적당한 전사 및 번역 조절 요소, 전사 인핸서 요소, 전사 종결자 등은 발현 벡터에서 사용될 수 있다 (예를 들면, U6 프로모터, H1 프로모터, 등; 상기 참고) (참고 예를 들면, Bitter 등 (1987) Methods in Enzymology, 153:516-544).
일부 구현예에서, DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드는 RNA로서 제공될 수 있다. 그와 같은 경우에, DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드를 인코딩하는 RNA는 직접적인 화학 합성에 의해 생산될 수 있거나 DNA-표적화 RNA를 인코딩하는 DNA로부터 시험관 내에서 전사될 수 있다. DNA 템플레이트로부터 RNA를 합성하는 방법은 대상체기술에 공지되어 있다. 일부 경우에서, DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드를 인코딩하는 RNA는 RNA 폴리머라제 효소 (예를 들면, T7 폴리머라제, T3 폴리머라제, SP6 폴리머라제, 등)을 사용하여 시험관 내에서 합성될 것이다. 일단 합성되면, RNA는 표적 DNA와 직접적으로 접촉할 수 있거나 핵산을 세포에 도입하는 공지된 기술 (예를 들면, 미세주입, 전기천공, 형질감염, 등) 중 임의의 것에 의해 세포에 도입될 수 있다.
(DNA 또는 RNA로서 도입된) DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 및/또는 부위 지향적 변형 폴리펩타이드 (DNA 또는 RNA로서 도입된) 및/또는 공여체 폴리뉴클레오타이드는 잘 개발된 형질감염 기술을 사용하여 세포에 제공될 수 있다; 참고, 예를 들면 Angel 및 Yanik (2010) PLoS ONE 5(7): e11756, 및 상업적으로 이용가능한 TransMessenger® 시약 (Qiagen), StemfectTM RNA 형질감염 키트 (Stemgent), 및 TransIT®-mRNA 형질감염 키트 (Mirus Bio LLC). 또한 하기를 참조한다: Beumer 등 (2008) Efficient gene targeting in Drosophila by direct embryo injection with zinc-finger nucleases. PNAS 105(50):19821-19826. 대안적으로, DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 키메라 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드를 인코딩하는 핵산은 DNA 벡터 상에 제공될 수 있다. 핵산을 표적 세포로 이동시키는데 유용한 많은 벡터, 예를 들면 플라스미드, 코스미드, 미니서클, 파아지, 바이러스, 등이 이용가능하다. 핵산(들)을 포함하는 벡터는 예를 들면 플라스미드, 미니서클 DNA, 바이러스 예컨대 사이토메갈로바이러스, 아데노바이러스, 등으로서 에피솜 상태로 유지될 수 있거나, 상동 재조합 또는 랜덤 통합, 예를 들면 레트로바이러스-유도된 벡터 예컨대 MMLV, HIV-1, ALV, 등을 통해 표적 세포 게놈에 통합될 수 있다.
벡터는 직접적으로 당해 세포에게 직접적으로 제공될 수 있다. 환언하면, 세포는 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 키메라 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드를 인코딩하는 핵산을 포함하는 벡터와 접촉되고 이로써 벡터는 세포에 의해 취해진다. 전기천공, 염화칼슘 형질감염, 미세주입, 및 리포펙션을 포함하는, 세포를 플라스미드인 핵산 벡터와 접촉하는 방법은 대상체기술에 공지되어 있다. 바이러스 벡터 전달에 대해, 세포는 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 키메라 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드. 레트로바이러스를 인코딩하는 핵산을 포함하는 바이러스 입자와 접촉되고, 예를 들면, 렌티바이러스는, 본 발명의 방법에 특이 적당하다. 통상적으로 사용된 레트로바이러스 벡터는 "불량성"이고, 즉 생산적인 감염에 필요한 바이러스 단백질을 생산할 수 없다. 오히려, 벡터의 복제는 패키징 세포주에서 성장을 필요로 한다. 관심 핵산을 포함하는 바이러스 입자를 산출하기 위해, 핵산을 포함하는 레트로바이러스 핵산은 패키징 세포주를 패키징하여 바이러스 캡시드에 포장된다. 상이한 패키징 세포주는 캡시드에 편입될 상이한 엔빌로프 단백질 (동종지향성, 암포트로픽 또는 친이종성)를 제공하고, 이러한 엔빌로프 단백질은 세포에 대한 바이러스 입자의 특이성을 결정한다 (쥣과 및 랫트에 대한 동종지향성; 인간, 개 및 마우스를 포함하는 대부분의 포유동물 세포형에 대한 암포트로픽; 및 쥣과 세포를 제외한 대부분의 포유동물 세포형에 대한 친이종성). 적절한 패키징 세포주는, 세포가 포장된 바이러스 입자에 의해 표적화되는 것을 보장하기 위해 사용될 수 있다. 재프로그래밍 인자를 인코딩하는 핵산을 포함하는 레트로바이러스 벡터를 패키징 세포주에 도입하고 패키징 계통에 의해 산출된 바이러스 입자를 수집하는 것을 포함하는 방법은 대상체기술에 공지되어 있다. 핵산은 직접적인 마이크로-주사 (예를 들면, RNA의 제브라피쉬 배아로의 주사)에 의해 또한 도입될 수 있다.
DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 키메라 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드를 인코딩하는 핵산을 당해 세포에게 제공하기 위해 사용된 벡터는 관심 핵산의 발현, 즉, 전사 활성화을 유도하기 위해 적당한 프로모터를 전형적으로 포함할 것이다. 환언하면, 관심 핵산은 프로모터에 작동가능하게 연결될 것이다. 도처에 작용 프로모터, 예를 들면, CMV-β-액틴 프로모터, 또는 유도성 프로모터, 예컨대 프로모터을 포함할 수 있고, 이들은 특정 세포 집단에서 활성이거나 약물 예컨대 테트라사이클린의 존재에 반응한다. 전사 활성화에 의해, 전사는 적어도 약 10 배까지, 적어도 약 100 배까지, 더 보통 적어도 약 1000 배까지 표적 세포에서 기저 수준 초과로 증가될 것으로 의도된다. 또한, DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 키메라 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드를 당해 세포에게 제공하기 위해 사용된 벡터는 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 키메라 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드를 취한 세포를 확인하기 위해, 표적 세포에서 선별 마커를 인코딩하는 핵산 서열을 포함할 수 있다.
당해 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 키메라 부위 지향적 변형 폴리펩타이드는 대신에 DNA와 접촉하기 위해 사용될 수 있거나 RNA로서 세포에 도입될 수 있다. RNA를 세포에 도입하는 방법은 대상체기술에 공지되어 있고, 예를 들면, 직접적인 주사, 형질감염, 또는 DNA의 도입을 위해 사용된 임의의 다른 방법을 포함할 수 있다.
당해 부위 지향적 변형 폴리펩타이드는 대신에 폴리펩타이드로서 세포에 제공될 수 있다. 그와 같은 폴리펩타이드는 생성물의 용해도를 증가시키는 폴리펩타이드 도메인에 임의로 융합될 수 있다. 도메인은 규정된 프로테아제 절단 부위, 예를 들면 TEV 프로테아제에 의해 절단된 TEV 서열을 통해 폴리펩타이드에 연결될 수 있다. 링커는 또한 1 이상의 가요성 서열, 예를 들면 1 내지 10 개의 글리신 잔기를 포함할 수 있다. 일부 구현예에서, 융합 단백질의 절단은 예를 들면 0.5 내지 2 M 우레아의 존재에서, 용해도 등을 증가시키는 폴리펩타이드 및/또는 폴리뉴클레오타이드의 존재에서, 생성물의 용해도를 유지하는 완충제에서 수행된다. 관심 도메인은 엔도솜분해적 도메인, 예를 들면 인플루엔자 HA 도메인; 및 생산을 돕는 다른 폴리펩타이드, 예를 들면 IF2 도메인, GST 도메인, GRPE 도메인, 등을 포함한다. 폴리펩타이드는 개선된 안정성을 위해 제형될 수 있다. 예를 들면, 펩타이드는 페길화될 수 있고, 여기서 폴리에틸렌옥시 그룹은 혈류에서 증대된 수명을 제공한다.
추가로 또는 대안적으로, 당해 부위 지향적 변형 폴리펩타이드는 세포에 의한 흡수를 촉진하기 위해 폴리펩타이드 투과하는 도메인에 융합될 수 있다. 수많은 투과하는 도메인은 대상체기술에 공지되어 있고 펩타이드, 펩타이도모사체, 및 비-펩타이드 담체를 포함하는, 본 발명의 비-통합 폴리펩타이드에서 사용될 수 있다. 예를 들면, 투과하는 펩타이드는 아미노산 서열 RQIKIWFQNRRMKWKK (서열번호://)을 포함하는, 페네트라틴으로 불리는 드로소필라 멜라노가스테르 전사 인자 안테나패디아의 제 3 알파 나사선구조로부터 유도될 수 있다. 또 하나의 예로서, 투과하는 펩타이드는 HIV-1 tat 염기성 영역 아미노산 서열을 포함하고, 이 서열은, 예를 들면, 자연-발생 tat 단백질의 아미노산 49-57을 포함할 수 있다. 다른 투과하는 도메인은 폴리-아르기닌 모티프, 예를 들면, HIV-1 rev 단백질, 노나-아르기닌, 옥타-아르기닌, 등의 아미노산 34-56의 영역을 포함한다. (참고, 예를 들면, Futaki 등 (2003) Curr Protein Pept Sci. 2003 Apr; 4(2): 87-9 및 446; 및 Wender 등 (2000) Proc. Natl. Acad. Sci. U.S.A 2000 Nov. 21; 97(24):13003-8; 공개된 미국 특허 출원 20030220334; 20030083256; 20030032593; 및 20030022831, 이들은 전좌 펩타이드 및 펩토이드의 교시에 대해 참조로 본원에 구체적으로 편입되어 있음). 노나-아르기닌 (R9) 서열은 특성화된 더 효율적인 PTD 중의 하나이다 (Wender 등 2000; Uemura 등 2002). 융합된 부위는 폴리펩타이드의 생물학적 활성, 분비 또는 결합 특성을 최적화하기 위해 선택될 수 있다. 최적의 부위는 일상적인 실험과정에 의해 결정될 것이다.
당해 부위 지향적 변형 폴리펩타이드는 시험관내 또는 진핵 세포 또는 원핵 세포에 의해 생산될 수 있고, 언폴딩, 예를 들면 열 변성, DTT 감소, 등에 의해 추가로 가공될 수 있고 당해분야에서 공지된 방법을 사용하여 추가로 재폴딩될 수 있다.
일차 서열을 변경시키지 않는 관심 변형은 폴리펩타이드의 화학 유도체화, 예를 들면, 아실화, 아세틸화, 카복실화, 아마이드화, 등을 포함한다. 당화의 변형, 예를 들면 그것의 합성 및 가공 동안에 폴리펩타이드의 당화 패턴을 변형시키거나 추가 처리 단계에서; 예를 들면 당화, 예컨대 포유동물 글리코실화 또는 탈글리코실화 효소에 영향을 주는 효소에 폴리펩타이드를 노출하여 만들어진 것들이 또한 포함된다. 인산화된 아미노산 잔기, 예를 들면 포스포티로신, 포스포세린, 또는 포스포트레오닌을 갖는 서열이 또한 포함된다.
단백분해 분해에 대한 그의 내성을 개선하고, 표적 서열 특이성을 변화시키고, 용해도 특성을 최적화하고, 단백질 활성 (예를 들면, 전사 조절 활성, 효소 활성, 등)을 변경하거나 치료제로서 더 적당하도록 하기 위해 보통 분자 생물학적 기술 및 합성 화학을 사용하여 변형되었던 DNA-표적화 RNA 및 부위 지향적 변형 폴리펩타이드가 본 발명에서 또한 포함된다. 그와 같은 폴리펩타이드의 유사체는 자연 발생 L-아미노산, 예를 들면 D-아미노산 또는 비-자연 발생 합성 아미노산 이외의 잔기를 함유하는 것들을 포함한다. D-아미노산은 아미노산 잔기의 일부 또는 모두 대신 치환될 수 있다.
부위 지향적 변형 폴리펩타이드는 대상체기술에서 공지된 종래의 방법을 사용하여 시험관내 합성으로 제조될 수 있다. 다양한 상업적 합성 장치는 이용가능한, 예를 들면, 자동화된 합성기 (Applied Biosystems, Inc., Beckman) 등이다. 합성기를 사용하여, 자연 발생 아미노산은 비천연 아미노산으로 치환될 수 있다. 제조의 특정한 순서 및 방식은 편리, 경제적, 순도 필요한, 등에 의해 결정될 것이다.
원한다면, 다양한 그룹은 합성 또는 발현 동안에 펩타이드로 도입될 수 있고, 이것은 다른 분자 또는 표면에의 연결을 허용한다. 따라서 시스테인은 티오에테르, 금속 착이온에 연결하기 위한 히스티딘, 아마이드 또는 에스테르를 형성하는 카복실 그룹, 아마이드를 형성하는 아미노 그룹, 등을 만들기 위해 사용될 수 있다.
부위 지향적 변형 폴리펩타이드는 재조합 합성의 종래의 방법에 따라 또한 단리 및 정제될 수 있다. 용해물은 발현 숙주를 제조할 수 있고 용해물은 HPLC, 배제 크로마토그래피, 겔 전기영동, 친화성 크로마토그래피, 또는 다른 정제 기술을 사용하여 정제된다. 대부분의 파트에 대해, 사용될 조성물은 생성물의 제조 및 그의 정제 방법과 관련된 오염물질과 관련하여 원하는 생성물의 적어도 20중량 %, 더 보통 적어도 약 75중량 %, 바람직하게는 적어도 약 95중량 %를 포함하고, 치료적 목적을 위해서는, 보통 적어도 약 99.5중량 %를 포함할 것이다. 보통, 백분율은 총 단백질을 기준으로 할 것이다.
DNA 절단 및 재조합, 또는 표적 DNA에 대한 임의의 원하는 변형, 또는 표적 DNA와 연관된 폴리펩타이드에 대한 임의의 원하는 변형을 유도하기 위해, DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드는, 핵산 또는 폴리펩타이드로서 도입되든지, 약 30 분 내지 약 24 시간, 예를 들면, 1 시간, 1.5 시간, 2 시간, 2.5 시간, 3 시간, 3.5 시간 4 시간, 5 시간, 6 시간, 7 시간, 8 시간, 12 시간, 16 시간, 18 시간, 20 시간, 또는 임의의 다른 기간 약 30 분 내지 약 24 시간 동안 세포에 제공되고, 이 시간은 약 매 1 일 내지 약 매 4 일, 예를 들면, 매 1.5 일, 매 2 일, 매 3 일, 또는 임의의 다른 빈도 약 매 1 일 내지 약 매 4 일의 빈도로 반복될 수 있다. 제제(들)은 당해 세포에게 1회 이상, 예를 들면 1 회, 2 회, 3 회, 또는 3 회 초과 제공될 수 있고, 세포는 각 접촉 이벤트 다음의 일부의 시간의 양 예를 들면 16-24 시간 동안 제제(들)과 함께 인큐베이션이 허용되고, 그 후 배지는 새로운 배지로 대체되고 세포는 추가로 배양된다.
2 이상의 상이한 표적 복합체가 세포에 제공되는 사례에서 (예를 들면, 동일 또는 상이한 표적 DNA 내의 상이한 서열에 대해 상보적인 2 개의 상이한 DNA-표적화 RNA), 복합체는 (예를 들면 2 개의 폴리펩타이드 및/또는 핵산으로서) 동시에 제공되거나, 동시에 전달될 수 있다. 대안적으로, 연속하여 제공될 수 있고, 예를 들면 표적화 복합물이 먼저 제공되고, 그 다음 제 2 표적화 복합물, 등이 제공되고, 또는 그 반대이다.
전형적으로, 효과적인 양의 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드는 절단을 유도하기 위해 표적 DNA 또는 세포에 제공된다. DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드의 효과적인 양은 음성 대조군, 예를 들면 빈 벡터 또는 무관한 폴리펩타이드와 접촉된 세포에 관하여 2 개의 상동 서열 사이에 관찰된 표적 변형의 양에서 2--배 증가 또는 그 초과를 유도하는 양이다. 즉, DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드의 효과적인 양 또는 용량은 표적 DNA 영역에서 관찰된 표적 변형의 양에서 2-배 증가, 3-배 증가, 4-배 증가 또는 그 초과, 일부 예에서 관찰된 재조합의 양 5-배 증가, 6-배 증가 또는 그 초과, 때때로 7-배 또는 8-배 증가 또는 그 초과, 예를 들면 관찰된 재조합의 양에서 10-배, 50-배, 또는 100-배 또는 그 초과의 증가, 일부 예에서, 200-배, 500-배, 700-배, 또는 1000-배 또는 그 초과의 증가, 예를 들면 5000-배, 또는 10,000-배 증가를 유도할 것이다. 표적 변형의 양은 임의의 편리한 방법에 의해 측정될 수 있다. 예를 들면, 재조합될 때, 활성 리포터를 인코딩하는 핵산을 구성할 반복 서열에 의해 플랭킹된 DNA-표적화 RNA의 표적 분절 (표적 서열)의 상보적 서열을 포함하는 침묵의 리포터 구조체는 세포로 공형질감염될 수 있고, 리포터 단백질의 양은 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드의 접촉한 후, 예를 들면 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드와 접촉한 후2 시간, 4 시간, 8 시간, 12 시간, 24 시간, 36 시간, 48 시간, 72 시간 또는 그 초과 동안 평가될 수 있다. 또 하나로서, 더 민감도 검정, 예를 들면, 표적 DNA 서열을 포함하는 관심 게놈 DNA 영역에서의 재조합의 정도는 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드의 접촉한 후, 예를 들면 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드와 접촉한 후 2 시간, 4 시간, 8 시간, 12 시간, 24 시간, 36 시간, 48 시간, 72 시간 또는 그 초과 동안 영역의 PCR 또는 서던 하이브리드화에 의해 평가될 수 있다.
세포를 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드와 접촉시키는 것은 임의의 배양 배지에서 및 세포의 생존을 촉진하는 임의의 배양 조건 하에서 일어날 수 있다. 예를 들면, 세포는 임의의 적절한 배양기에서 현탁될 수 있고, 상기 배양기는, 소태아 혈청 또는 열 불활성화된 염소 혈청 (약 5-10%), L-글루타민 티올, 특히 2-머캅토에탄올, 및 항생제, 예를 들면 페니실린 및 스트렙토마이신으로 보강된 편리한, 예컨대 이스코브 변형된 DMEM 또는 RPMI 1640. 배양물은 세포가 반응성인 성장 인자를 함유할 수 있다. 성장 인자는, 본원에서 규정된 바와 같이, 막통과 수용체에 대한 특이적 효과를 통해 배양물에서 또는 온전한 조직에서 세포의 생존, 성장 및/또는 분화를 촉진할 수 있는 분자이다. 성장 인자는 폴리펩타이드 및 비-폴리펩타이드 인자를 포함한다. 세포의 생존을 촉진하는 조건은 비상동 말단 연결 및 상동성-유도된 치유가 전형적으로 허용된다.
폴리뉴클레오타이드 서열을 표적 DNA 서열에 삽입하는 것이 바람직한 적용에서, 삽입될 공여체 서열을 포함하는 폴리뉴클레오타이드는 세포에 의해 또한 제공된다. "공여체 서열" 또는 "공여체 폴리뉴클레오타이드"란, 절단 부위에서 삽입될 핵산 서열이 부위 지향적 변형 폴리펩타이드에 의해 유도된다는 것을 의미한다. 공여체 폴리뉴클레오타이드는, 그것과 상동성을 보유하는 게놈 서열과 사이의 상동성-유도된 치유를 지지하기 위해 예를 들면 절단 부위의 약 50 개 이하의 염기 내에서, 예를 들면 약 30 개 염기, 약 15 개 염기, 약 10 개 염기, 약 5 염기 내에서 절단 부위에서 게놈 서열에 대해 충분한 상동성, 예를 들면 절단 부위를 플랭킹하거나, 절단 부위를 즉시 플랭킹하는 뉴클레오타이드 서열과의 70%, 80%, 85%, 90%, 95%, 또는 100% 상동성을 함유할 것이다. 공여체와 게놈 서열 사이의 서열 상동성 (또는 10 내지 200 개의 뉴클레오타이드, 또는 그 초과의 임의의 적분 값)의 대략 25, 50, 100, 또는 200 개의 뉴클레오타이드, 또는 200 개 초과의 뉴클레오타이드는 상동성-유도된 치유를 지지할 것이다. 공여체 서열은 임의의 길이, 예를 들면 10 개의 뉴클레오타이드 또는 그 초과, 50 개의 뉴클레오타이드 또는 그 초과, 100 개의 뉴클레오타이드 또는 그 초과, 250 개의 뉴클레오타이드 또는 그 초과, 500 개의 뉴클레오타이드 또는 그 초과, 1000 개의 뉴클레오타이드 또는 그 초과, 5000 개의 뉴클레오타이드 또는 그 초과, 등일 수 있다.
공여체 서열은 대체되는 게놈 서열과 전형적으로 동일하지 않다. 오히려, 공여체 서열은, 충분한 상동성이 상동성-유도된 치유를 지지하기 위해 존재하는 한 게놈 서열에 대해 적어도 1 이상의 단일 염기 변화, 삽입, 결실, 반전 또는 재배열을 함유할 수 있다. 일부 구현예에서, 공여체 서열은 상동성의 2 개의 영역에 의해 플랭킹된 비-상동 서열을 포함하고, 이로써 표적 DNA 영역과 2 개의 플랭킹 서열 사이의 상동성-유도된 치유로 표적 영역에서 비-상동 서열이 삽입된다. 공여체 서열은 관심 DNA 영역에 대해 상동성이 아니고 관심 DNA 영역으로 삽입에 대해 의도되지 않는 서열을 함유하는 벡터 골격을 또한 포함할 수 있다. 일반적으로, 공여체 서열의 상동 영역(들)은, 재조합을 원하는 게놈 서열과 적어도 50% 서열 동일성을 가질 것이다. 어떤 구현예에서, 60%, 70%, 80%, 90%, 95%, 98%, 99%, 또는 99.9% 서열 동일성이 존재한다. 1% 내지 100% 서열 동일성의 임의의 값은 공여체 폴리뉴클레오타이드의 길이에 의존하여 존재할 수 있다.
공여체 서열은 절단 부위에서 공여체 서열의 성공적인 삽입에 대해 평가하기 위해 사용될 수 있거나 일부 경우에서 (예를 들면, 표적화된 게놈 유전자좌위에서 발현을 나타내기 위한) 다른 목적을 위해 사용될 수 있는 게놈 서열, 예를 들면 제한 부위, 뉴클레오타이드 다형성, 선별 마커 (예를 들면, 약물 내성 유전자, 형광 단백질, 효소 등), 등과 비교된 바와 같이 어떤 서열 차이를 포함할 수 있다. 일부 경우에서, 코딩 영역에서 위치하면, 그와 같은 뉴클레오타이드 서열 차이는 아미노산 서열을 변경하지 않을 것이거나, 침묵의 아미노산 변화 (즉, 단백질의 구조 또는 기능에 영향을 미치지 않는 변화)를 만들 수 있을 것이다. 대안적으로, 이들 서열 차이는 마커 서열의 제거 동안에 늦게 활성화될 수 있는 플랭킹 재조합 서열 예컨대 FLPs, loxP 서열, 등을 포함할 수 있다.
공여체 서열은 단일가닥 DNA, 단일가닥 RNA, 이중-가닥 DNA, 또는 이중-가닥 RNA로서 세포에 제공될 수 있다. 선형 또는 원형 형태로 세포에 도입될 수 있다. 선형 형태로 도입되면, 공여체 서열의 말단은은 당해분야의 숙련가에게 공지된 방법에 의해 (예를 들면, 핵산외부분해 분해로부터) 보호될 수 있다. 예를 들면, 1 이상의 디데옥시뉴클레오타이드 잔기는 선형 분자의 3' 말단에 부가되고/거나 자가-상보적 올리고뉴클레오타이드는 하나 또는 둘 모두 말단에 결찰된다. 참고, 예를 들면, Chang 등 (1987) Proc. Natl. Acad Sci USA 84:4959-4963; Nehls 등 (1996) Science 272:886-889. 외인성 폴리뉴클레오타이드를 분해로부터 보호하는 추가의 방법은, 비제한적으로, 말단 아미노 그룹(들)의 부가 및 변형된 뉴클레오타이드간 연결 예컨대, 포스포로티오에이트, 포스포르아미데이트, 및 O-메틸 리보오스 또는 데옥시리보스 잔기의 사용을 포함한다. 선형 공여체 서열의 말단을 보호하는 대안으로서, 서열의 추가의 길이는 재조합에 영향을 주지 않으면서 분해될 수 있는 상동성의 영역 외부에서 포함될 수 있다. 공여체 서열은 추가의 서열 예컨대, 항생제 내성을 인코딩하는 유전자, 복제 기원 및 프로모터를 갖는 벡터 분자의 일부로서 세포에 도입될 수 있다. 게다가, 공여체 서열은 기존의 핵산으로서, 제제 예컨대 리포좀 또는 폴록사머와 복합된 핵산으로서 도입될 수 있거나, DNA-표적화 RNA를 인코딩하는 핵산 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드에 대해 상기에서 기재된 바와 같이 바이러스 (예를 들면, 아데노바이러스, AAV)에 의해 전달될 수 있다.
상기에서 기재된 방법에 따라, 관심 DNA 영역은 절단 및 변형될 수 있고, 즉 생체외에서 "유전적으로 변형된다". 일부 구현예에서, 선별 마커가 관심 DNA 영역에 도입되었을 때와 같이, 세포의 모집단은 유전적으로 변형된 세포를 잔여 모집단으로부터 분리하여 유전적 변형을 포함하는 것에 대해 풍부할 수 있다. 풍부해지기 전에, "유전적으로 변형된" 세포는 단지 세포성 모집단의 약 1% 또는 그 초과 (예를 들면, 2% 또는 그 초과, 3% 또는 그 초과, 4% 또는 그 초과, 5% 또는 그 초과, 6% 또는 그 초과, 7% 또는 그 초과, 8% 또는 그 초과, 9% 또는 그 초과, 10% 또는 그 초과, 15% 또는 그 초과, 또는 20% 또는 그 초과)를 이룰 수 있다. "유전적으로 변형된" 세포의 분리는 사용된 선별 마커에 적절한 임의의 편리한 분리 기술에 의해 달성될 수 있다. 예를 들면, 형광 마커가 삽입되었다면, 세포는 형광 활성화된 세포 분류에 의해 분리될 수 있고, 반면에 세포 표면 마커가 삽입되었다면, 세포는 친화성 분리 기술, 예를 들면 자기성 분리, 친화성 크로마토그래피, 고체 매트릭스에 부착된 친화성 시약에 의한 "패닝(panning)", 또는 다른 편리한 기술에 의해 이질적인 모집단으로부터 분리될 수 있다. 정확한 분리를 제공하는 기술은 정교화의 가변도, 예컨대 다중 칼라 채널, 저각도 및 둔각 광 산란 검출 채널, 임피던스 채널, 등을 갖는 형광 활성화된 세포 정렬기를 포함한다. 세포는 죽은 세포와 연관된 염료 (예를 들면 프로피듐 아이오다이드)를 이용하여 죽인 세포에 대항하여 선택될 수 있다. 유전적으로 변형된 세포의 생존력에 지나치게 해롭지 않은 임의의 기술이 이용될 수 있다. 변형된 DNA을 포함하는 세포에 대해 크게 풍분한 세포 조성물은 이러한 방식으로 달성된다. "크게 풍부한"이란, 유전적으로 변형된 세포가 세포 조성물의 70% 또는 그 초과, 75% 또는 그 초과, 80% 또는 그 초과, 85% 또는 그 초과, 90% 또는 그 초과, 예를 들면, 세포 조성물의 약 95% 또는 그 초과, 또는 98% 또는 그 초과일 것이라는 것을 의미한다. 환언하면, 조성물은 유전적으로 변형된 세포의 실질적으로 순수한 조성물일 수 있다.
본원에서 기재된 방법에 의해 생산된 유전적으로 변형된 세포는 즉시 사용될 수 있다. 대안적으로, 세포는 액체 질소 온도에서 냉동될 수 있고 장기간 동안 저장될 수 있고, 녹고 재사용될 수 있다. 그와 같은 경우에, 세포는 그와 같은 냉동 온도에서 세포를 보존하기 위해 대상체기술에서 통상적으로 사용된 바와 같이 10% 디메틸설폭사이드 (DMSO), 50% 혈청, 40% 완충된 배지, 또는 일부 다른 그와 같은 용액에서 보통 냉동될 것이고, 냉동된 배양 세포를 용해하기 위해 대상체기술에서 통상적으로 공지된 바와 같이 녹는다.
유전적으로 변형된 세포는 다양한 배양 조건 하에서 시험관 내에서 배양될 수 있다. 세포는 배양물에서 확장될 수 있고, 즉 그의 증식을 촉진하는 조건하에서 성장될 수 있다. 배양 배지는, 예를 들면 한천, 메틸셀룰로오스, 등을 함유하는 액체 또는 반-고체일 수 있다. 세포 집단은 소태아 혈청 (약 5-10%), L-글루타민 티올, 특히 2-머캅토에탄올, 및 항생제, 예를 들면 페니실린 및 스트렙토마이신으로 보통 보강된 적절한 배양기, 예컨대 이스코브 변형된 DMEM 또는 RPMI 1640에서 현탁될 수 있다. 배양물은, 조절 T 세포가 반응성인 성장 인자를 함유할 수 있다. 성장 인자는, 본원에서 규정된 바와 같이, 막통과 수용체에 대한 특이적 효과를 통해 배양물 또는 온전한 조직에서 세포의 생존, 성장 및/또는 분화를 촉진할 수 있는 분자이다. 성장 인자는 폴리펩타이드 및 비-폴리펩타이드 인자를 포함한다.
이런식으로 유전적으로 변형되었던 세포는 예를 들면 질환을 치료하기 위한 유전자 요법과 같은 목적으로 또는 항바이러스로서, 항병원체, 또는 항암 치료, 농업에서 유전적으로 변형된 유기체의 생산을 위해, 또는 생물학적 연구를 위해 대상체에 이식될 수 있다. 대상체는 신생아, 유년성, 또는 성인일 수 있다. 포유동물 대상체가 특정한 관심 대상이다. 당해 방법으로 치료될 수 있는 포유동물 종은 갯과 및 고양이; 말; 소; 양; 등 및 영장류, 특히 인간을 포함한다. 동물 모형, 특히 작은 포유동물 (예를 들면 마우스, 랫트, 기니아 피그, 햄스터, 토끼목 (예를 들면, 토끼), 등)은 실험실 조사를 위해 사용될 수 있다.
세포는, 예를 들면 이식되고 있는 조직에서 그의 성장 및/또는 조직화을 지지하기 위해 상기 대상체에게 단독으로 또는 적당한 기질 또는 매트릭스와 함께 제공될 수 있다. 보통, 적어도 1x103 개의 세포, 예를 들면 5x103 개의 세포, 1x104 개의 세포, 5x104 개의 세포, 1x105 개의 세포, 1 x 106 개의 세포 또는 그 초과가 투여될 것이다. 세포는 하기의 경로 중 임의의 것을 통해 상기 대상체에게 도입될 수 있다: 비경구, 피하, 정맥내, 두개내, 척수내, 안구내, 또는 척수액으로. 세포는 주사로, 카테터, 등으로 도입될 수 있다. 국소 전달, 즉, 부상의 부위에의 전달을 위한 방법의 예는, 예를 들면 하기를 포함한다: 예를 들면 척추강내 전달 동안 옴마야 저장기를 통해, (참고 예를 들면 US 특허 번호 5,222,982 및 5385582, 이는 본원에 참조로 편입되어 있음); 볼러스 주사에 의해, 예를 들면 주사기에 의해, 예를 들면 관절에; 연속적 주입에 의해, 예를 들면 캐뉼라삽입에 의해, 예를 들면 대류로 (참고 예를 들면 US 출원 번호 20070254842, 이는 본원에 참조로 편입되어 있음); 또는 세포가 반대로 부착된 디바이스를 이식하여 (참고 예를 들면 US 출원 번호 20080081064 및 20090196903, 이는 본원에 참조로 편입되어 있음). 세포는 이식유전자 동물 (예를 들면, 이식유전자 마우스)를 산출하기 위해 목적으로 배아 (예를 들면, 주머니배)에 또한 도입될 수 있다.
대상체에 대한 치료의 투여의 수는 변할 수 있다. 유전적으로 변형된 세포를 상기 대상체에게 투여하는 것은 1회용 이벤트일 수 있고; 그러나 어떤 상황에서, 그와 같은 치료는 제한된 기강 동안 개선을 유도할 수 있고 반복된 치료의 진행 중인 시리즈를 필요로 한다. 다른 상황에서, 유전적으로 변형된 세포의 다중 투여는, 효과가 관찰되기 전에 필요할 수 있다. 정확한 프로토콜은 질환 또는 병태, 질환의 단계 및 치료될 개별적인 대상체의 파라미터에 의존한다.
본 발명의 어떤 다른 측면에서, DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드는 세포성 DNA를 생체내에서 변형시키기 위해, 또한 유전자 요법, 예를 들면 질환을 치료하기 위한 유전자 요법과 같은 목적을 위해 또는 항바이러스로서, 항병원체, 또는 항암 치료, 농업에서 유전적으로 변형된 유기체의 생산을 위해, 또는 생물학적 연구를 위해 이용된다. 이들 생체내 구현예에서, DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드는 개체에 직접적으로 투여된다. DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드는 펩타이드, 소분자 및 핵산의 대상체에의 투여를 위해 대상체기술에서의 임의의 수의 공지된 방법에 의해 투여될 수 있다. DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드는 다양한 제형에 편입될 수 있다. 더 상세하게는, 본 발명의 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드는 적절한 약제학적으로 허용가능한 담체 또는 희석제와 조합하여 약제학적 조성물로 제형될 수 있다.
약제학적 제제는 약제학적으로 허용가능한 비히클에 존재하는 하나 이상의 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드을 포함하는 조성물이다. "약제학적으로 허용가능한 비히클"은 연방 또는 주 정부의 관리 기관에 의해 승인되거나 포유동물, 예컨대 인간에서 사용하기 위한 미국 약전 또는 다른 일반적으로 인식된 약전에 열거된 비히클일 수 있다. 용어 "비히클"은 본 발명의 화합물이 포유동물에의 투여를 위해 제형된 희석제, 아쥬반트, 부형제, 또는 담체를 의미한다. 그와 같은 약제학적 비히클은 하기일 수 있다: 지질, 예를 들면 리포좀, 예를 들면 리포좀 덴드리머; 석유, 동물, 식물성 또는 합성 기원, 예컨대 땅콩 오일, 대두 오일, 미네랄 오일, 참께 오일 등, 염수의 것을 포함하는 액체, 예컨대 물 및 오일; 아카시아검, 젤라틴, 전분 페이스트, 탈크, 케라틴, 콜로이드 실리카, 우레아, 등. 또한, 보조제, 안정화제, 증점제, 윤활제 및 착색제가 사용될 수 있다. 약제학적 조성물은 고체, 반-고체, 액체 또는 가스성 형태, 예컨대 정제, 캡슐, 분말, 과립, 연고, 용액, 좌약, 주사, 흡입제, 겔, 마이크로구형체, 및 에어로졸에서 제제로 제형될 수 있다. 이와 같이, DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드의 투여는 다양한 방식으로 달성될 수 있고, 그 투여는 경구, 구강, 직장, 비경구, 복강내, 진피내, 경피, 기관내, 안구내, 등, 투여를 포함한다. 활성제는 투여 후 전신일 수 있거나 영역 투여의 사용, 점막내 투여, 또는 이식의 부위에서 활성 용량을 유지하도록 작용하는 임플란트의 사용에 의해 국재화될 수 있다. 활성제는 즉각적인 활성을 위해 제형될 수 있거나 지속 방출용으로 제형될 수 있다.
일부 병태, 특히 중추신경계 병태에 대해, 혈액-뇌 장벽 (BBB)를 가로지르는 제제를 제형하는 것이 필요할 수 있다. 혈액-뇌 장벽 (BBB)을 통한 약물 전달의 하나의 전략은 삼투 수단 예컨대 만니톨 또는 류코트리엔에 의해, 또는 생화학적으로 혈관활성 물질 예컨대 브라디키닌의 사용에 의해 BBB의 파손을 수반한다. 뇌종양에 대한 특이한 제제를 표적으로 하기 위해 BBB 개구를 사용하기 위한 잠재력은 또흔 선택이다. BBB 파괴 약물은, 조성물이 혈관내 주사에 의해 투여될 때 본 발명의 치료 조성물과 공-투여될 수 있다. BBB를 통과하기 위한 다른 전략은 하기를 포함하는 내인성 수송 시스템의 사용을 수반할 수 있다: 카베올린-1 매개된 통과세포외배출, 담체-매개된 수송체 예컨대 글루코오스 및 아미노산 담체, 인슐린에 대한 수용체-매개된 통과세포외배출 또는 트랜스페린, 및 활성 유출 수송체 예컨대 p-당단백질. 활성 수송 모이어티는 혈관의 내피 벽을 가로지르는 수송을 촉진하기 위해 본 발명에서 사용하기 위한 치료 화합물에 또한 접합될 수 있다. 대안적으로, BBB 뒤에 있는 치료제의 약물 전달은 하기에 의해서 일 수 있다: 예를 들면 옴마야 저장기를 통해 국소 전달, 예를 들면 척추강내 전달에 의해 (참고 예를 들면 US 특허 번호 5,222,982 및 5385582, 이는 본원에 참조로 편입되어 있음); 볼러스 주사에 의해, 예를 들면 주사기에 의해, 예를 들면 초자체내로 또는 두개내로; 연속적 주입에 의해, 예를 들면 대류에 의한 캐뉼라삽입에 의해 (참고 예를 들면 US 출원 번호 20070254842, 이는 본원에 참조로 편입되어 있음); 또는 제제가 반대로 부착되었던 디바이스를 이식하여 (참고 예를 들면 US 출원 번호 20080081064 및 20090196903, 이는 본원에 참조로 편입되어 있음).
전형적으로, 효과적인 양의 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드가 제공된다. 생체외 방법에 대해 상기에서 논의된 바와 같이, 생체 내의 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드의 효과적인 양 또는 효과적인 용량은 음성 대조군, 예를 들면 빈 벡터 또는 무관한 폴리펩타이드와 접촉된 세포에 대해 2 개의 상동 서열 사이에 관찰된 재조합의 양에서 2 배 증가 또는 그 초과를 유도하기 위한 양이다. 재조합의 양은 예를 들면 상기에서 기재되고 대상체기술에서 공지된 바와 같이 임의의 편리한 방법에 의해 측정될 수 있다. 투여될 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드의효과적인 양 또는 효과적인 용량의 계산은 당해분야의 숙련가의 기술 내에 있고, 당해분야의 숙련가에게 일상적인 것이다. 투여될 최종 양은 투여 경로 및 치료될 장애 또는 병태의 본성에 대해 의존할 것이다
특정한 환자에게 주어질 치료 조성물의 효과적인 양은 다양한 인자에 의존할 것이고, 그 몇 개는 환자에 따라 상이한 것이다. 능숙한 임상의는, 질환 상태의 요구에 따라 진행을 멈추거나 역전시키기 위해 환자에게 투여하기 위한 치료제의 효과적인 양을 결정할 수 있을 것이다. LD50 동물 데이타, 및 제제를 위해 이용가능한 다른 정보를 이용하여, 임상의는 투여 경로에 따라 개체에 대한 최대 안전한 용량을 결정할 수 있다. 예를 들면, 정맥내로 투여된 용량은 치료 조성물이 투여되는 유체의 더 큰 신체를 고려하면 척추강내로 투여된 용량보다 더 많을 것이다. 유사하게, 신체로부터 빠르게 없어지는 조성물은 치료적 농도를 유지하기 위해 고용량으로, 또는 반복된 용량으로 투여될 수 있다. 보통 기술을 이용하여, 능숙한 임상의는 일상적인 임상시험의 과정에서 특정한 치료제의 복용량을 최적화할 것이다.
약제에 포함하기 위해, DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드는 적당한 상업적 공급원으로부터 수득될 수 있다. 일반적인 제의로서, 용량 당 비경구로 투여된 총 약제학적으로 효과적인 양의 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드는 용량 반응 곡선에 의해 측정될 수 있는 범위일 것이다.
DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드를 기반으로 하는 치료제, 즉 치료적 투여를 위해 사용될 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드의 제제는, 멸균해야 한다. 멸균은 멸균한 여과 막 (예를 들면, 0.2 μm 막)을 통한 여과에 의해 쉽게 달성된다. 치료 조성물은 일반적으로, 멸균한 접근 포트를 갖는 용기, 예를 들면, 정맥내 용액 백 또는 피하 주사 바늘로 뚫을 수 있는 스토퍼를 갖는 바이알에 놓여진다. DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드 및/또는 공여체 폴리뉴클레오타이드를 기반으로 하는 치료제는 수용액으로서 또는 재구성용 동결건조된 제형으로서 장치 또는 다중-용량 용기, 예를 들면, 밀봉된 앰풀 또는 바이알에서 저장될 수 있다. 동결건조된 제형의 예로서, 10-mL 바이알은 화합물의 5 ml의 멸균한-여과된 1% (w/v) 수용액으로 충전되고 수득한 혼합물은 동결건조된다. 수액은 주사용 정균 수를 사용하여 동결건조된 화합물을 재구성하여 제조된다.
약제학적 조성물은, 원하는 제형에 따라, 동물 또는 인간 투여용 약제학적 조성물을 제형하기 위해 통상적으로 사용되는 비히클로서 규정된 희석제의 약제학적으로-허용가능한, 비-독성 담체를 포함할 수 있다. 희석제는 조합의 생물학적 활성에 영향을 주지 않도록 선택된다. 그와 같은 희석제의 예는 증류수, 완충된 물, 생리적 염수, PBS, 링거액, 덱스트로오스 용액, 및 한스 용액이다. 또한, 약제학적 조성물 또는 제형는 다른 담체, 아쥬반트, 또는 비-독성, 비치료제, 비면역원성 안정제, 부형제 등을 포함할 수 있다. 성물은 대략적인 생리적 조건에 대한 추가의 물질, 예컨대 pH 조정제 및 완충제, 독성 조정제, 습윤제 및 세제를 또한 포함할 수 있다.
조성물은 임의의 다양한 안정제, 예컨대 항산화제를 또한 포함할 수 있다. 약제학적 조성물이 폴리펩타이드를 포함할 때, 폴리펩타이드는 폴리펩타이드의 생체내 안정성을 증가시키거나, 달리 그것의 약리적 특성를 향상시키고 (예를 들면, 폴리펩타이드의 반감기를 증가시키고, 그것의 독성을 감소시키고, 용해도 또는 흡수를 향상시키는) 다양한 공지된 화합물과 복합될 수 있다. 그와 같은 변형 또는 착화제의 예는 설페이트, 글루코네이트, 시트레이트 및 포스페이트를 포함한다. 조성물의 핵산 또는 폴리펩타이드는 그의 생체내 속성을 향상시키는 분자와 또한 복합될 수 있다. 그와 같은 분자는, 예를 들면, 탄수화물, 폴리아민, 아미노산, 다른 펩타이드, 이온 (예를 들면, 나트륨, 칼륨, 칼슘, 마그네슘, 망간), 및 지질을 포함한다.
다양한 유형의 투여에 적당한 제형에 관한 추가 안내는 하기에서 발견될 수 있다: Remington's Pharmaceutical Sciences, Mace Publishing Company, Philadelphia, Pa., 17th ed. (1985). 약물 전달의 방법의 간단한 검토에 대해, Langer, Science 249:1527-1533 (1990)를 참고한다.
약제학적 조성물은 예방적 및/또는 치료적 처리를 위해 투여될 수 있다. 활성 성분의 독성 및 치료적 효능은 예를 들면, LD50 (모집단의 50%에 대해 치명적인 용량) 및 ED50 (모집단의 50%에서 치료 효과적인 용량)을 결정하는 것을 포함하는 세포 배양 및/또는 실험 동물에서 표준 약제학적 절차에 따라 결정될 수 있다. 독성과 치료 효과 사이의 용량 비는 치료 지수이고 비 LD50/ED50로서 표현될 수 있다. 큰 치료적 지수를 나타내는 치료제가 바람직하다.
세포 배양 및/또는 동물 연구로부터 수득된 데이타는 인간을 위한 복용량의 범위를 제형하는데 사용될 수 있다. 활성 성분의 복용량은 전형적으로 저독성을 갖는 ED50를 포함하는 순환 농도의 범위 내에 있다. 복용량은 이용된 복용 형태 및 이용된 투여 경로에 의존하는 이러한 범위 내에서 변할 수 있다.
약제학적 조성물을 제형하기 위해 사용된 성분은 바람직하게는 고순도이고 잠재적으로 유해한 오염물질이 실질적으로 없다 (예를 들면, 적어도 National Food (NF) 등급, 일반적으로 적어도 분석적 등급, 및 더욱 전형적으로 적어도 약품 등급). 게다가, 생체내 용도로 의도된 조성물은 보통 멸균한다. 주어진 화합물이 사용 전에 합성되어야 하는 정도로, 수득한 생성물은 전형적으로, 임의의 잠재적으로 독성제, 특히 합성 또는 정제 공정 동안에 존재할 수 있는 임의의 내독소가 실질적으로 없다. 모 투여용 조성물은 또한 멸균하고, 실질적으로 등장이고 GMP 조건 하에서 만들어진다.
특정한 환자에게 주어질 치료 조성물의 효과적인 양은 다양한 인자에 의존할 것이고, 그 몇 개는 환자에 따라 상이한 것이다. 능숙한 임상의는, 질환 상태의 요구에 따라 진행을 멈추거나 역전시키기 위해 환자에게 투여하기 위한 치료제의 효과적인 양을 결정할 수 있을 것이다. LD50 동물 데이타, 및 제제에 대해 이용가능한 다른 정보를 이용하여, 임상의는 투여 경로에 따라 개인의 최대 안전한 용량을 결정할 수 있다. 예를 들면, 정맥내로 투여된 용량은, 치료 조성물이 투여되고 있는 유체의 더 큰 신체를 고려할 때 척추강내로 투여된 용량보다 더 많을 수 있다. 유사하게, 신체로부터 빠르게 없어지는 조성물은 치료적 농도를 유지하기 위해 고용량으로, 또는 반복된 용량으로 투여될 수 있다. 보통 기술을 이용하여, 능숙한 임상의는 일상적인 임상시험의 과정에서 특정한 치료제의 복용량을 최적화할 수 있을 것이다.
유전적으로 변형된 숙주세포
당해 개시내용은 단리된 유전적으로 변형된 숙주세포를 포함하는 유전적으로 변형된 숙주세포를 제공하고, 여기서 당해 유전적으로 변형된 숙주세포는 하기를 포함한다 (하기로 유전적으로 변형되었다): 1) 외인성 DNA-표적화 RNA; 2) 외인성 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산; 3) 외인성 부위 지향적 변형 폴리펩타이드 (예를 들면, 자연 발생 Cas9; 변형된, 즉, 돌연변이된 또는 변이체, Cas9; 키메라 Cas9; 등); 4) 부위 지향적 변형 폴리펩타이드를 인코딩하는 외인성 뉴클레오타이드 서열을 포함하는 핵산; 또는 5) 상기의 임의의 조합. 당해 유전적으로 변형된 세포는 숙주세포를 하기 예의 것으로 유전적으로 변형하여 산출된다: 1) 외인성 DNA-표적화 RNA; 2) 외인성 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산; 3) 외인성 부위 지향적 변형 폴리펩타이드; 4) 부위 지향적 변형 폴리펩타이드를 인코딩하는 외인성 뉴클레오타이드 서열을 포함하는 핵산; 또는 5) 상기의 임의의 조합).
표적 세포로 적당한 모든 세포는 또한 유전적으로 변형된 숙주세포인 것에도 적당하다. 예를 들면, 관심 유전적으로 변형된 숙주세포는 하기일 수 있다: 임의의 유기체로부터의 세포 (예를 들면 박테리아 세포, 고세균 세포, 단일-세포 진핵 유기체의 세포, 식물 세포, 조류 세포, 예를 들면, 보트라이오코쿠스 브라우니이 , 클 라마이도모나스 레인하르드티이 , 난노클로롭시스 가디타나 , 클로렐라 파이레노이도사, 사르가섬 파텐스 C. 아가르드, 등, 진균 세포 (예를 들면, 효모 세포), 동물 세포, 무척추동물 동물 (예를 들면 초파리, 자포동물, 극피동물, 선충, 등)로부터의 세포, 척추동물 동물 (예를 들면, 물고기, 양서류, 파충류, 새, 포유동물)로부터의 세포, 포유동물로부터의 세포 (예를 들면, 돼지, 소, 염소, 양, 설치류, 랫트, 마우스, 비-인간 영장류, 인간, 등), 등.
일부 구현예에서, 유전적으로 변형된 숙주세포는 부위 지향적 변형 폴리펩타이드 (예를 들면, 자연 발생 Cas9; 변형된, 즉, 돌연변이된 또는 변이체, Cas9; 키메라 Cas9; 등)를 인코딩하는 외인성 뉴클레오타이드 서열을 포함하는 핵산으로 유전적으로 변형되었다. 유전적으로 변형된 숙주세포의 DNA는 세포에 DNA-표적화 RNA (또는 변형될 게놈 위치/서열을 결정할 DNA-표적화 RNA를 인코딩하는 DNA) 및 임의로 공여체 핵산을 도입하여 변형을 위해 표적화될 수 있다. 일부 구현예에서, 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열은 유도성 프로모터 (예를 들면, 열충격 프로모터, 테트라사이클린-조절된 프로모터, 스테로이드-조절된 프로모터, 금속-조절된 프로모터, 에스트로겐 수용체-조절된 프로모터, 등)에 작동가능하게 연결된다. 일부 구현예에서, 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열은 공간적으로 제한된 및/또는 일시적으로 제한된 프로모터 (예를 들면, 조직 특이적 프로모터, 세포형 특이적 프로모터, 등)에 작동가능하게 연결된다. 일부 구현예에서, 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열은 항시적 프로모터에 작동가능하게 연결된다.
일부 구현예에서, 당해 유전적으로 변형된 숙주세포는 시험관 내에 있다. 일부 구현예에서, 당해 유전적으로 변형된 숙주세포는 생체 내에 있다. 일부 구현예에서, 당해 유전적으로 변형된 숙주세포는 원핵 세포이거나 원핵 세포로부터 유도된다. 일부 구현예에서, 당해 유전적으로 변형된 숙주세포는 박테리아 세포이거나 박테리아 세포로부터 유도된다. 일부 구현예에서, 당해 유전적으로 변형된 숙주세포는 고세균 세포이거나 고세균 세포로부터 유도된다. 일부 구현예에서, 당해 유전적으로 변형된 숙주세포는 진핵 세포이거나 진핵 세포로부터 유도된다. 일부 구현예에서, 당해 유전적으로 변형된 숙주세포는 식물 세포이거나 식물 세포로부터 유도된다. 일부 구현예에서, 당해 유전적으로 변형된 숙주세포는 동물 세포이거나 동물 세포로부터 유도된다. 일부 구현예에서, 당해 유전적으로 변형된 숙주세포는 무척추동물 세포이거나 무척추동물 세포로부터 유도된다. 일부 구현예에서, 당해 유전적으로 변형된 숙주세포는 척추동물 세포이거나 척추동물 세포로부터 유도된다. 일부 구현예에서, 당해 유전적으로 변형된 숙주세포는 포유동물 세포이거나 포유동물 세포로부터 유도된다. 일부 구현예에서, 당해 유전적으로 변형된 숙주세포는 설치류 세포이거나 설치류 세포로부터 유도된다. 일부 구현예에서, 당해 유전적으로 변형된 숙주세포는 인간 세포이거나 인간 세포로부터 유도된다.
당해 개시내용은 추가로, 당해 유전적으로 변형된 세포의 자손을 제공하고, 상기 자손은 유도된 당해 유전적으로 변형된 세포로서 동일한 외인성 핵산 또는 폴리펩타이드를 포함할 수 있다. 당해 개시내용은 추가로, 당해 유전적으로 변형된 숙주세포를 포함하는 조성물을 제공한다.
유전적으로 변형된 줄기세포 및 유전적으로 변형된 전구 세포
일부 구현예에서, 당해 유전적으로 변형된 숙주세포는 유전적으로 변형된 줄기세포 또는 전구 세포. 적당한 숙주세포는, 예를 들면, 줄기세포 (성체 줄기 세포, 배아 줄기 세포, iPS 세포, 등) 및 전구 세포 (예를 들면, 심장 전구 세포, 신경 전구 세포, 등)을 포함한다. 적당한 숙주세포는 포유동물 줄기세포 및 전구 세포를 포함하고, 이들 세포는, 예를 들면, 설치류 줄기세포, 설치류 전구 세포, 인간 줄기세포, 인간 전구 세포 등을 포함한다. 적당한 숙주세포는 시험관내 숙주세포, 예를 들면, 단리된 숙주세포를 포함한다.
일부 구현예에서, 당해 유전적으로 변형된 숙주세포는 외인성 DNA-표적화 RNA 핵산을 포함한다. 일부 구현예에서, 당해 유전적으로 변형된 숙주세포는 외인성 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산을 포함한다. 일부 구현예에서, 당해 유전적으로 변형된 숙주세포는 외인성 부위 지향적 변형 폴리펩타이드 (예를 들면, 자연 발생 Cas9; 변형된, 즉, 돌연변이된 또는 변이체, Cas9; 키메라 Cas9; 등)을 포함한다. 일부 구현예에서, 당해 유전적으로 변형된 숙주세포는 부위 지향적 변형 폴리펩타이드를 인코딩하는 외인성 뉴클레오타이드 서열을 포함하는 핵산을 포함한다. 일부 구현예에서, 당해 유전적으로 변형된 숙주세포는 하기를 인코딩하는 외인성 뉴클레오타이드 서열을 포함하는 핵산을 포함한다: 1) DNA-표적화 RNA 및 2) 부위 지향적 변형 폴리펩타이드.
일부 경우에서, 부위 지향적 변형 폴리펩타이드는 아미노산 서열을 포함하고, 이 서열은 도 3에서 묘사된 Cas9/Csn1 아미노산 서열의 아미노산 7-166 또는 731-1003과, 또는 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 상응하는 부분과 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 99%, 또는 100%, 아미노산 서열 동일성을 갖는다.
조성물
본 발명은 당해 DNA-표적화 RNA 및/또는 부위 지향적 변형 폴리펩타이드를 포함하는 조성물을 제공한다. 일부 경우에서, 부위 지향적 변형 폴리펩타이드는 당해 키메라 폴리펩타이드이다. 당해 조성물은 당해 개시내용의 방법, 예를 들면, 표적 DNA의 부위 특이적인 변형 방법; 표적 DNA와 연관된 폴리펩타이드의 부위 특이적인 변형 방법; 등을 수행하는데 유용하다.
DNA - 표적화 RNA 를 포함하는 조성물
본 발명은 당해 DNA-표적화 RNA를 포함하는 조성물을 제공한다. 당해 조성물은, DNA-표적화 RNA에 추가하여, 하기 중 하나 이상을 포함할 수 있다: 염, 예를 들면, NaCl, MgCl2, KCl, MgSO4, 등; 완충제, 예를 들면, Tris 완충제, N-(2-하이드록시에틸)피페라진-N'-(2-에탄설폰산) (HEPES), 2-(N-모폴리노)에탄설폰산 (MES), MES 나트륨 염, 3-(N-모폴리노)프로판설폰산 (MOPS), N-트리[하이드록시메틸]메틸 1-3-아미노프로판설폰산 (TAPS), 등; 가용화제; 세제, 예를 들면, 비-이온성 세제 예컨대 Tween-20, 등; 뉴클레아제 억제제; 등. 예를 들면, 일부 경우에서, 당해 조성물은 당해 DNA-표적화 RNA 및 핵산을 안정화시키는 완충제를 포함한다.
일부 구현예에서, 당해 조성물에 존재하는 DNA-표적화 RNA는 순수하고, 예를 들면, 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 98%, 적어도 약 99%, 또는 99% 초과 순수하고, 여기서 "순도 %"는 DNA-표적화 RNA가 다른 거대분자, 또는 DNA-표적화 RNA의 생산 동안에 존재할 수 있는 오염물질이 없는 인용된 퍼센트이라는 것을 의미한다.
당해 키메라 폴리펩타이드를 포함하는 조성물
본 발명은 당해 키메라 폴리펩타이드를 포함하는 조성물을 제공한다. 당해 조성물은, DNA-표적화 RNA에 추가하여, 하기 중 하나 이상을 포함할 수 있다: 염, 예를 들면, NaCl, MgCl2, KCl, MgSO4, 등; 완충제, 예를 들면, Tris 완충제, HEPES, MES, MES 나트륨 염, MOPS, TAPS, 등; 가용화제; 세제, 예를 들면, 비-이온성 세제 예컨대 Tween-20, 등; 프로테아제 억제제; 환원제 (예를 들면, 디티오트레이톨); 등.
일부 구현예에서, 당해 조성물에 존재하는 당해 키메라 폴리펩타이드는 순수하고, 예를 들면, 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 98%, 적어도 약 99%, 또는 99% 초과 순수하고, 여기서 "순도 %"는 부위 지향적 변형 폴리펩타이드가 다른 단백질, 다른 거대분자, 또는 키메라 폴리펩타이드의 생산 동안에 존재할 수 있는 오염물질이 없는 인용된 퍼센트인 것을 의미한다.
DNA - 표적화 RNA 및 부위 지향적 변형 폴리펩타이드를 포함하는 조성물
본 발명은 하기를 포함하는 조성물을 제공한다: (i) DNA-표적화 RNA 또는 그것을 인코딩하는 DNA 폴리뉴클레오타이드; 및 ii) 부위 지향적 변형 폴리펩타이드, 또는 그것을 인코딩하는 폴리뉴클레오타이드. 일부 경우에서, 부위 지향적 변형 폴리펩타이드는 당해 키메라 부위 지향적 변형 폴리펩타이드이다. 다른 사례에서, 부위 지향적 변형 폴리펩타이드는 자연-발생 부위 지향적 변형 폴리펩타이드이다. 일부 예에서, 부위 지향적 변형 폴리펩타이드는 표적 DNA를 변형시키는 효소 활성을 나타낸다. 다른 사례에서, 부위 지향적 변형 폴리펩타이드는 표적 DNA와 연관된 폴리펩타이드를 변형시키는 효소 활성을 나타낸다. 또 다른 사례에서, 부위 지향적 변형 폴리펩타이드는 표적 DNA의 전사를 조절한다.
본 발명은 하기를 포함하는 조성물을 제공한다: (i) 하기를 포함하는 상기에서 기재된 바와 같은 DNA-표적화 RNA, 또는 그것을 인코딩하는 DNA 폴리뉴클레오타이드: (a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및 (b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절; 및 (ii) 하기를 포함하는 부위 지향적 변형 폴리펩타이드, 또는 그것을 인코딩하는 폴리뉴클레오타이드: (a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및 (b) 부위 지향적 효소 활성을 나타내는 활성부로서, 상기 효소 활성의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부.
일부 예에서, 당해 조성물은 하기를 포함하는 조성물을 포함한다: (i) 하기를 포함하는 당해 DNA-표적화 RNA: (a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및 (b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절; 및 (ii) 하기를 포함하는 상기 부위 지향적 변형 폴리펩타이드: (a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및 (b) 부위 지향적 효소 활성을 나타내는 활성부로서, 상기 효소 활성의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부.
다른 구현예에서, 당해 조성물은 하기를 포함한다: (i) 하기를 포함하는 당해 DNA-표적화 RNA를 인코딩하는 폴리뉴클레오타이드: (a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및 (b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절; 및 (ii) 하기를 포함하는 부위 지향적 변형 폴리펩타이드를 인코딩하는 폴리뉴클레오타이드: (a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및 (b) 부위 지향적 효소 활성을 나타내는 활성부로서, 상기 효소 활성의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부.
일부 구현예에서, 당해 조성물은 이중-분자 DNA-표적화 RNA의 RNA 분자 둘 모두를 포함한다. 이와 같이, 일부 구현예에서, 당해 조성물은 타겟터-RNA의 듀플렉스-형성 분절에 대해 상보적인 듀플렉스-형성 분절을 포함하는 활성제-RNA를 포함한다 (참고 도 1a). 활성제-RNA 및 타겟터-RNA의 듀플렉스-형성 분절은 혼성화되어 DNA-표적화 RNA의 단백질-결합 세그먼트의 dsRNA 듀플렉스를 형성한다. 타겟터-RNA는 추가로, DNA-표적화 RNA의 DNA-표적화 분절 (단일가닥)을 제공하고 따라서 표적 DNA 내의 특이적 서열에 대한 DNA-표적화 RNA를 표적으로 한다. 하나의 비제한적인 예로서, 활성제-RNA의 듀플렉스-형성 분절은 뉴클레오타이드 서열을 포함하고, 이 서열은 서열 5'-UAGCAAGUUAAAAU-3' (서열번호:562)과 적어도 약 70%, 적어도 약 80%, 적어도 약 90%, 적어도 약 95%, 적어도 약 98%, 또는 100% 동일성을 갖는다. 또 하나의 비-제한적인 예로서, 타겟터-RNA의 듀플렉스-형성 분절은 뉴클레오타이드 서열을 포함하고, 이 서열은 서열 5'-GUUUUAGAGCUA-3' (서열번호:679)과 적어도 약 70%, 적어도 약 80%, 적어도 약 90%, 적어도 약 95%, 적어도 약 98%, 또는 100% 동일성을 갖는다.
당해 개시내용은 하기를 포함하는 조성물을 제공한다: (i) 하기를 포함하는 DNA-표적화 RNA, 또는 이것을 인코딩하는 DNA 폴리뉴클레오타이드: (a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및 (b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절; 및 (ii) 하기를 포함하는 부위 지향적 변형 폴리펩타이드, 또는 그것을 인코딩하는 폴리뉴클레오타이드: (a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및 (b) 표적 DNA 내에서 전사를 조절하는 활성부로서, 상기 표적 DNA 내의 조절된 전사의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부.
예를 들면, 일부 경우에서, 당해 조성물은 하기를 포함한다: (i) 하기를 포함하는 DNA-표적화 RNA: (a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및 (b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절; 및 (ii) 하기를 포함하는 상기 부위 지향적 변형 폴리펩타이드: (a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및 (b) 표적 DNA 내에서 전사를 조절하는 활성부로서, 상기 표적 DNA 내의 조절된 전사의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부.
또 하나의 예로서, 일부 경우에서, 당해 조성물은 하기를 포함한다: (i) 하기를 포함하는 DNA-표적화 RNA를 인코딩하는 폴리뉴클레오타이드: (a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및 (b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절; 및 (ii) 하기를 포함하는 부위 지향적 변형 폴리펩타이드를 인코딩하는 폴리뉴클레오타이드: (a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및 (b) 표적 DNA 내에서 전사를 조절하는 활성부로서, 상기 표적 DNA 내의 조절된 전사의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부.
당해 조성물은, i) 당해 DNA-표적화 RNA, 또는 이것을 인코딩하는 DNA 폴리뉴클레오타이드; 및 ii) 부위 지향적 변형 폴리펩타이드, 또는 그것을 인코딩하는 폴리뉴클레오타이드에 추가하여, 하기 중 하나 이상을 포함할 수 있다: 염, 예를 들면, NaCl, MgCl2, KCl, MgSO4, 등; 완충제, 예를 들면, Tris 완충제, HEPES, MES, MES 나트륨 염, MOPS, TAPS, 등; 가용화제; 세제, 예를 들면, 비-이온성 세제 예컨대 Tween-20, 등; 프로테아제 억제제; 환원제 (예를 들면, 디티오트레이톨); 등.
일부 경우에서, 조성물의 성분은 개별적으로 순수하고, 예를 들면, 각각의 성분은 적어도 약 75%, 적어도 약 80%, 적어도 약 90%, 적어도 약 95%, 적어도 약 98%, 적어도 약 99%, 또는 적어도 99%, 순수하다. 일부 경우에서, 당해 조성물의 개별 성분은 조성물에 부가되기 전에 순수하다.
예를 들면, 일부 구현예에서, 당해 조성물에 존재하는 부위 지향적 변형 폴리펩타이드는 순수하고, 예를 들면, 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 98%, 적어도 약 99%, 또는 99% 초과 순수하고, 여기서 "순도 %"는 부위 지향적 변형 폴리펩타이드가 다른 단백질 (예를 들면, 부위 지향적 변형 폴리펩타이드 이외의 단백질), 다른 거대분자, 또는 부위 지향적 변형 폴리펩타이드의 생산 동안에 존재할 수 있는 오염물질이 없는 인용된 퍼센트인 것을 의미한다.
키트
당해 개시내용은 당해 방법을 수행하기 위한 키트를 제공한다. 당해 키트는 하기: 부위 지향적 변형 폴리펩타이드; 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드를 포함하는 핵산; DNA-표적화 RNA; DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산; 활성제-RNA; 활성제-RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산; 타겟터-RNA; 및 타겟터-RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산. 부위 지향적 변형 폴리펩타이드; 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드를 포함하는 핵산; DNA-표적화 RNA; DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산; 활성제-RNA; 활성제-RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산; 타겟터-RNA; 및 타겟터-RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산 중 하나 이상을 포함할 수 있고, 상기에 상세히 기재되어 있다. 키트는 하기 중 2 이상을 포함하는 복합물을 포함할 수 있다: 부위 지향적 변형 폴리펩타이드; 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드를 포함하는 핵산; DNA-표적화 RNA; DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산; 활성제-RNA; 활성제-RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산; 타겟터-RNA; 및 타겟터-RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산.
일부 구현예에서, 당해 키트는 부위 지향적 변형 폴리펩타이드, 또는 그것을 인코딩하는 폴리뉴클레오타이드를 포함한다. 일부 구현예에서, 부위 지향적 변형 폴리펩타이드는 하기를 포함한다: (a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및 (b) 표적 DNA 내에서 전사를 조절하는 활성부로서, 상기 표적 DNA 내의 조절된 전사의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부. 일부 경우에서, 부위 지향적 변형 폴리펩타이드의 활성부는 감소된 또는 불활성화된 뉴클레아제 활성을 나타낸다. 일부 경우에서, 부위 지향적 변형 폴리펩타이드는 키메라 부위 지향적 변형 폴리펩타이드이다.
일부 구현예에서, 당해 키트는 하기를 포함한다: 부위 지향적 변형 폴리펩타이드, 또는 그것을 인코딩하는 폴리뉴클레오타이드, 및 부위 지향적 변형 폴리펩타이드를 재구성 및/또는 희석하기 위한 시약. 다른 구현예에서, 당해 키트는 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드를 포함하는 핵산 (예를 들면, DNA, RNA)을 포함한다. 일부 구현예에서, 당해 키트는 하기를 포함한다: 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드를 포함하는 핵산 (예를 들면, DNA, RNA); 및 부위 지향적 변형 폴리펩타이드를 재구성 및/또는 희석하기 위한 시약.
부위 지향적 변형 폴리펩타이드, 또는 그것을 인코딩하는 폴리뉴클레오타이드를 포함하는 당해 키트는, 1 이상 추가의 시약을 추가로 포함할 수 있고, 여기서 그와 같은 추가의 시약은 하기로부터 선택될 수 있다: 부위 지향적 변형 폴리펩타이드를 세포에 도입하기 위한 완충제; 세척 완충제; 대조군 시약; 대조군 발현 벡터 또는 RNA 폴리뉴클레오타이드; DNA로부터 부위 지향적 변형 폴리펩타이드의 시험관내 생산을 위한 시약, 등. 일부 경우에서, 당해 키트에서 포함된 부위 지향적 변형 폴리펩타이드는 상기에서 기재된 바와 같이 키메라 부위 지향적 변형 폴리펩타이드이다.
일부 구현예에서, 당해 키트는 하기를 포함하는 DNA-표적화 RNA, 또는 이것을 인코딩하는 DNA 폴리뉴클레오타이드를 포함한다: (a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및 (b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절. 일부 구현예에서, DNA-표적화 RNA는 추가로, (상기에서 기재된 바와 같은) 제 3 분절을 포함한다. 일부 구현예에서, 당해 키트는 하기를 포함한다: (i) 하기를 포함하는 DNA-표적화 RNA, 또는 이것을 인코딩하는 DNA 폴리뉴클레오타이드: (a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및 (b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절; 및 (ii) 부위 지향적 변형 폴리펩타이드, 또는 그것을 인코딩하는 폴리뉴클레오타이드, 부위 지향적 변형 폴리펩타이드는 하기를 포함한다: (a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및 (b) 부위 지향적 효소 활성을 나타내는 활성부로서, 상기 효소 활성의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부. 일부 구현예에서, 부위 지향적 변형 폴리펩타이드의 활성부는 효소 활성 (예를 들면, 돌연변이를 통해 불활성화된 뉴클레아제를 포함하는) 효소 활성을 나타내지 않는다. 일부 경우에서, 키트는 DNA-표적화 RNA 및 부위 지향적 변형 폴리펩타이드를 포함한다. 다른 사례에서, 키트는 하기를 포함한다: (i) DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산; 및 (ii) 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산.
또 하나의 예로서, 당해 키트는 하기를 포함할 수 있다: (i) 하기를 포함하는, DNA-표적화 RNA, 또는 이것을 인코딩하는 DNA 폴리뉴클레오타이드: (a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및 (b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절; 및 (ii) 하기를 포함하는 상기 부위 지향적 변형 폴리펩타이드, 또는 그것을 인코딩하는 폴리뉴클레오타이드: (a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및 (b) 표적 DNA 내에서 전사를 조절하는 활성부로서, 상기 표적 DNA 내의 조절된 전사의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부. 일부 경우에서, 키트는 하기를 포함한다: (i) DNA-표적화 RNA; 및 부위 지향적 변형 폴리펩타이드. 다른 사례에서, 키트는 하기를 포함한다: (i) DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산; 및 (ii) 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산.
당해 개시내용은 하기를 포함하는 키트를 제공한다: (1) 하기를 포함하는 재조합 발현 벡터: (i) 하기를 포함하는 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열: (a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및 (b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절; 및 (ii) 하기를 포함하는 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열: (a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및 (b) 부위 지향적 효소 활성을 나타내는 활성부로서, 상기 효소 활성의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부; 및 (2) 상기 발현 벡터의 재구성 및/또는 희석을 위한 시약.
당해 개시내용은 하기를 포함하는 키트를 제공한다: (1) 하기를 포함하는 재조합 발현 벡터: (i) 하기를 포함하는 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열: (a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및 (b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절; 및 (ii) 하기를 포함하는 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열: (a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및 (b) 표적 DNA 내에서 전사를 조절하는 활성부로서, 상기 표적 DNA 내의 조절된 전사의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부; 및 (2) 재조합 발현 벡터의 재구성 및/또는 희석을 위한 시약.
당해 개시내용은 하기를 포함하는 키트를 제공한다: (1) 하기를 포함하는 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산을 포함하는 재조합 발현 벡터: (i) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및 (ii) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절; 및 (2) 재조합 발현 벡터의 재구성 및/또는 희석을 위한 시약. 이러한 키트의 일부 구현예에서, 키트는 하기를 포함하는 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 재조합 발현 벡터를 포함한다: (a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및 (b) 부위 지향적 효소 활성을 나타내는 활성부로서, 상기 효소 활성의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부. 이러한 키트의 다른 구현예에서, 키트는 하기를 포함하는 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 재조합 발현 벡터를 포함한다: (a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및 (b) 표적 DNA 내에서 전사를 조절하는 활성부로서, 상기 표적 DNA 내의 조절된 전사의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부.
상기 키트의 임의의 것의 일부 구현예에서, 키트는 활성제-RNA 또는 타겟터-RNA를 포함한다. 상기 키트의 임의의 것의 일부 구현예에서, 키트는 단일-분자 DNA-표적화 RNA를 포함한다. 상기 키트의 임의의 것의 일부 구현예에서, 키트는 2 이상의 이중-분자 또는 단일-분자 DNA-표적화 RNA를 포함한다. 상기 키트의 임의의 것의 일부 구현예에서, (예를 들면, 2 이상의 DNA-표적화 RNA를 포함하는) DNA-표적화 RNA는 어레이 (예를 들면, RNA 분자의 어레이, DNA-표적화 RNA(들)를 인코딩하는 DNA 분자의 어레이, 등)으로서 제공될 수 있다. 그와 같은 키트는, 예를 들면, 당해 부위 지향적 변형 폴리펩타이드를 포함하는 상기 기재된 유전적으로 변형된 숙주세포와 함께 사용하는데 유용할 수 있다. 상기 키트의 임의의 것의 일부 구현예에서, 키트는 추가로, 원하는 유전적 변형에 영향을 주기 위해 공여체 폴리뉴클레오타이드를 포함한다. 당해 키트의 성분은 별개의 용기 내에 있을 수 있거나; 단일 용기에서 조합될 수 있다.
상기-기재된 키트의 임의의 것은 1 이상의 추가의 시약을 추가로 포함할 수 있고, 여기서 그와 같은 추가의 시약은 하기로부터 선택될 수 있다: 희석 완충제; 재구성 용액; 세척 완충제; 대조군 시약; 대조군 발현 벡터 또는 RNA 폴리뉴클레오타이드; DNA로부터 부위 지향적 변형 폴리펩타이드의 시험관내 생산을 위한 시약, 등.
상기-언급된 성분 외에, 당해 키트는 당해 방법을 실시하기 위해 키트의 성분을 사용하기 위한 성분을 추가로 포함할 수 있다. 당해 방법을 실시하는 설명은 적당한 기록 매체 상에 일반적으로 기록된다. 예를 들면, 설명은 기재, 예컨대 종이 또는 플라스틱, 등 상에 인쇄될 수 있다. 이와 같이, 설명은 키트 또는 그의 성분의 용기의 라벨링 (즉, 패키징 또는 서브패키징과 연관된) 키트 또는 그의 성분의 용기의 라벨링 등에서 포장내 삽입물로서 키트 내에 존재할 수 있다. 다른 구현예에서, 설명은 적당한 컴퓨터 판독가능한 저장 매체, 예를 들면 CD-ROM, 디스켓, 플래시 드라이브, 등 상에 존재하는 전자 저장 데이터로서 존재한다. 또 다른 구현예에서, 실제 설명은 키트 내에 존재하지 않지만, 예를 들면 인터넷을 통해 떨어져 있는 공급원으로부터의 설명을 얻기 위한 수단이 제공된다. 이러한 구현예의 예는, 설명이 보여질 수 있고/거나 설명이 다운로드된 웹 어드레서를 포함하는 키트이다. 설명으로서, 설명을 얻기 위한 이러한 수단은 적당한 기재 상에 기록된다.
비-인간 유전적으로 변형된 유기체
일부 구현예에서, 유전적으로 변형된 숙주세포는 부위 지향적 변형 폴리펩타이드 (예를 들면, 자연 발생 Cas9; 변형된, 즉, 돌연변이된 또는 변이체, Cas9; 키메라 Cas9; 등)를 인코딩하는 외인성 뉴클레오타이드 서열을 포함하는 핵산으로 유전적으로 변형되었다. 그와 같은 세포가 진핵 단일-세포 유기체이면, 이때 변형된 세포는 유전적으로 변형된 유기체로 간주될 수 있다. 일부 구현예에서, 당해 비-인간 유전적으로 변형된 유기체는 Cas9 이식유전자 다중세포 유기체이다.
일부 구현예에서, 당해 유전적으로 변형된 비-인간 숙주세포 (예를 들면, 부위 지향적 변형 폴리펩타이드, 예를 들면, 자연 발생 Cas9; 변형된, 즉, 돌연변이된 또는 변이체, Cas9; 키메라 Cas9; 등을 인코딩하는 외인성 뉴클레오타이드 서열을 포함하는 핵산으로 유전적으로 변형된 세포)는 당해 유전적으로 변형된 비-인간 유기체 (예를 들면, 마우스, 물고기, 개구리, 파리, 벌레, 등)을 산출할 수 있다. 예를 들면, 유전적으로 변형된 숙주세포가 만능 줄기세포 (즉, PSC) 또는 생식세포 (예를 들면, 정자, 난모세포, 등)이면, 전체 유전적으로 변형된 유기체는 유전적으로 변형된 숙주세포로부터 유도될 수 있다. 일부 구현예에서, 유전적으로 변형된 숙주세포는 유전적으로 변형된 유기체를 생기게 할 수 있는 생체내 또는 시험관내에서 만능 줄기세포 (예를 들면, ESC, iPSC, 만능 식물 줄기세포, 등) 또는 생식세포 (예를 들면, 정자, 난모세포, 등)이다. 일부 구현예에서 유전적으로 변형된 숙주세포는 척추동물 PSC (예를 들면, ESC, iPSC, 등)이고, (예를 들면 비-키메라/비-모자이크 유전적으로 변형된 유기체를 산출하기 위해 교미된 키메라/모자이크 동물을 산출하기 위해 PSC를 주머니배에 주입하여; 식물의 경우에 그라프팅에 의해; 등) 유전적으로 변형된 유기체를 산출하기 위해 사용된다. 본원에서 기재된 방법을 포함하는, 유전적으로 변형된 유기체를 생산하기 위한 임의의 편리한 방법/프로토콜은 부위 지향적 변형 폴리펩타이드 (예를 들면, 자연 발생 Cas9; 변형된, 즉, 돌연변이된 또는 변이체, Cas9; 키메라 Cas9; 등)를 인코딩하는 외인성 뉴클레오타이드 서열을 포함하는 핵산을 포함한는 유전적으로 변형된 숙주세포를 생산하는데 적당하다. 유전적으로 변형된 유기체를 생산하는 방법은 대상체기술에 공지되어 있다. 예를 들면, 참고 Cho 등, Curr Protoc Cell Biol. 2009 Mar;Chapter 19:Unit 19.11: Generation of transgenic mice; Gama 등, Brain Struct Funct. 2010 Mar;214(2-3):91-109. Epub 2009 Nov 25: Animal transgenesis: an overview; Husaini 등, GM Crops. 2011 Jun-Dec;2(3):150-62. Epub 2011 Jun 1: Approaches for gene targeting and targeted gene expression in plants.
일부 구현예에서, 유전적으로 변형된 유기체는 본 발명의 방법을 위한 표적 세포를 포함하고, 따라서 표적 세포용 공급원으로서 간주될 수 있다. 예를 들면, 부위 지향적 변형 폴리펩타이드 (예를 들면, 자연 발생 Cas9; 변형된, 즉, 돌연변이된 또는 변이체, Cas9; 키메라 Cas9; 등)를 인코딩하는 외인성 뉴클레오타이드 서열을 포함하는 핵산을 포함하는 유전적으로 변형된 세포가 유전적으로 변형된 유기체를 산출하기 위해 사용된다면, 이때 유전적으로 변형된 유기체의 세포는 부위 지향적 변형 폴리펩타이드 (예를 들면, 자연 발생 Cas9; 변형된, 즉, 돌연변이된 또는 변이체, Cas9; 키메라 Cas9; 등)를 인코딩하는 외인성 뉴클레오타이드 서열을 포함하는 핵산을 포함한다. 그와 같은 일부 구현예에서, 세포 또는 유전적으로 변형된 유기체의 세포의 DNA는 세포 또는 세포들에 DNA-표적화 RNA (또는 DNA-표적화 RNA를 인코딩하는 DNA) 및 임의로 공여체 핵산을 도입하여 변형을 위해 표적화될 수 있다. 예를 들면, DNA-표적화 RNA (또는 DNA-표적화 RNA를 인코딩하는 DNA)의 유전적으로 변형된 유기체의 세포의 서브셋 (예를 들면, 뇌 세포, 창자 세포, 신장 세포, 폐 세포, 혈구, 등)에의 도입은, 그의 게놈 위치가 도입된 DNA-표적화 RNA의 DNA-표적화 서열에 따른 것인 변형을 위한 그와 같은 세포의 DNA를 표적으로 할 수 있다.
일부 구현예에서, 유전적으로 변형된 유기체는 본 발명의 방법을 위한 표적 세포의 공급원이다. 예를 들면, 부위 지향적 변형 폴리펩타이드 (예를 들면, 자연 발생 Cas9; 변형된, 즉, 돌연변이된 또는 변이체, Cas9; 키메라 Cas9; 등)를 인코딩하는 외인성 뉴클레오타이드 서열을 포함하는 핵산으로 유전적으로 변형된 세포를 포함하는 은 유전적으로 변형된 유기체는 유전적으로 변형된 세포, 예를 들면 PSC (예를 들면, ESCs, iPSC, 정자, 난모세포, 등), 뉴런, 전구 세포, 심근세포, 등의 공급원을 제공할 수 있다.
일부 구현예에서, 유전적으로 변형된 세포는 부위 지향적 변형 폴리펩타이드 (예를 들면, 자연 발생 Cas9; 변형된, 즉, 돌연변이된 또는 변이체, Cas9; 키메라 Cas9; 등)를 인코딩하는 외인성 뉴클레오타이드 서열을 포함하는 핵산을 포함하는 PSC이다. 이와 같이, 상기 PSC는 표적 세포일 수 있고 이로써 PSC의 DNA는 상기 PSC에 DNA-표적화 RNA (또는 DNA-표적화 RNA를 인코딩하는 DNA) 및 임의로 공여체 핵산을 도입하여 변형을 위해 표적화될 수 있고, 변형의 게놈 위치는 도입된 DNA-표적화 RNA의 DNA-표적화 서열을 따를 것이다. 따라서, 일부 구현예에서, 본원에서 기재된 방법은 당해 유전적으로 변형된 유기체로부터 유도된 PSC의 DNA를 변형시키고 (예를 들면, 임의의 원하는 게놈 위치를 결실시키고/거나 대체하기 위해 사용될 수 있다). 그와 같은 변형된 PSC는 그때, 하기 둘 모두를 갖는 유기체를 산출하기 위해 사용될 수 있다: (i) 부위 지향적 변형 폴리펩타이드 (예를 들면, 자연 발생 Cas9; 변형된, 즉, 돌연변이된 또는 변이체, Cas9; 키메라 Cas9; 등)를 인코딩하는 외인성 뉴클레오타이드 서열을 포함하는 핵산 및 (ii) PSC에 도입되었던 DNA 변형.
부위 지향적 변형 폴리펩타이드 (예를 들면, 자연 발생 Cas9; 변형된, 즉, 돌연변이된 또는 변이체, Cas9; 키메라 Cas9; 등)를 인코딩하는 외인성 뉴클레오타이드 서열을 포함하는 핵산은 (예를 들면, 핵산이 무작위로 숙주세포 게놈에 통합될 때) 미공지된 프로모터의 조절하에 있을 수 있거나 (즉, 그것에 작동가능하게 연결될 수 있고), 공지된 프로모터의 조절 하에 있을 수 있다 (즉, 그것에 작동가능하게 연결될 수 있다). 적당한 공지된 프로모터는 임의의 공지된 프로모터일 수 있고 항시적으로 활성인 프로모터 (예를 들면, CMV 프로모터), 유도성 프로모터 (예를 들면, 열충격 프로모터, 테트라사이클린-조절된 프로모터, 스테로이드-조절된 프로모터, 금속-조절된 프로모터, 에스트로겐 수용체-조절된 프로모터, 등), 공간적으로 제한된 및/또는 일시적으로 제한된 프로모터 (예를 들면, 조직 특이적 프로모터, 세포형 특이적 프로모터, 등), 등을 포함한다.
당해 유전적으로 변형된 유기체 (예를 들면 그의 세포가 부위 지향적 변형 폴리펩타이드, 예를 들면, 자연 발생 Cas9; 변형된, 즉, 돌연변이된 또는 변이체, Cas9; 키메라 Cas9; 등를 인코딩하는 뉴클레오타이드 서열을 포함하는 유기체)는 하기의 예를 포함하는 임의의 유기체일 수 있다: 식물; 조류; 무척추동물 (예를 들면, 자포동물, 극피동물, 벌레, 파리, 등); 척추동물 (예를 들면, 물고기 (예를 들면, 제브라피쉬, 복어, 금붕어, 등), 양서류 (예를 들면, 도롱룡, 개구리, 등), 파충류, 새, 포유동물, 등); 유제류 (예를 들면, 염소, 돼지, 양, 소, 등); 설치류 (예를 들면, 마우스, 랫트, 햄스터, 기니아 피그); 토끼목 (예를 들면, 토끼); 등.
일부 경우에서, 부위 지향적 변형 폴리펩타이드는 아미노산 서열을 포함하고, 이 서열은 도 3에서 묘사된 Cas9/Csn1 아미노산 서열의 아미노산 7-166 또는 731-1003과, 또는 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 상응하는 부분과 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 99%, 또는 100%, 아미노산 서열 동일성을 갖는다.
이식유전자 비-인간 동물
상기에서 기재된 바와 같이, 일부 구현예에서, 당해 핵산 (예를 들면, 부위 지향적 변형 폴리펩타이드, 예를 들면, 자연 발생 Cas9; 변형된, 즉, 돌연변이된 또는 변이체, Cas9; 키메라 Cas9; 등)를 인코딩하는 뉴클레오타이드 서열 또는 당해 재조합 발현 벡터는 부위 지향적 변형 폴리펩타이드를 생산하는 이식유전자 동물을 산출하기 위해 이식유전자로서 사용된다. 따라서, 본 발명은 추가로, 이식유전자 비-인간 동물을 제공하고, 이 동물은 상기에서 기재된 바와 같이 부위 지향적 변형 폴리펩타이드, 예를 들면, 자연 발생 Cas9; 변형된, 즉, 돌연변이된 또는 변이체, Cas9; 키메라 Cas9; 등을 인코딩하는 당해 뉴클레오타이드 서열을 포함하는 핵산을 포함하는 이식유전자를 포함한다. 일부 구현예에서, 이식유전자 비-인간 동물의 게놈은 당해 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함한다. 일부 구현예에서, 이식유전자 비-인간 동물은 유전적 변형에 대한 동종접합이다. 일부 구현예에서, 이식유전자 비-인간 동물은 유전적 변형에 대한 이종접합니다. 일부 구현예에서, 이식유전자 비-인간 동물은 하기이다: 척추동물, 예를 들면, 물고기 (예를 들면, 제브라피쉬, 금붕어, 복어, 동굴어, 등), 양서류 (개구리, 도롱룡, 등), 새 (예를 들면, 닭, 칠면조, 등), 파충류 (예를 들면, 뱀, 도마뱀, 등), 포유동물 (예를 들면, 유제류, 예를 들면, 돼지, 소, 염소, 양, 등; 토끼목 (예를 들면, 토끼); 설치류 (예를 들면, 랫트, 마우스); 비-인간 영장류; 등), 등.
부위 지향적 변형 폴리펩타이드 (예를 들면, 자연 발생 Cas9; 변형된, 즉, 돌연변이된 또는 변이체, Cas9; 키메라 Cas9; 등)를 인코딩하는 외인성 뉴클레오타이드 서열을 포함하는 핵산은 (예를 들면, 핵산이 무작위로 숙주세포 게놈에 통합될 때) 미공지된 프로모터의 조절하에 있을 수 있거나 (즉, 그것에 작동가능하게 연결될 수 있고), 공지된 프로모터의 조절 하에 있을 수 있다 (즉, 그것에 작동가능하게 연결될 수 있다). 적당한 공지된 프로모터는 임의의 공지된 프로모터일 수 있고 하기를 포함한다: 항시적으로 활성인 프로모터 (예를 들면, CMV 프로모터), 유도성 프로모터 (예를 들면, 열충격 프로모터, 테트라사이클린-조절된 프로모터, 스테로이드-조절된 프로모터, 금속-조절된 프로모터, 에스트로겐 수용체-조절된 프로모터, 등), 공간적으로 제한된 및/또는 일시적으로 제한된 프로모터 (예를 들면, 조직 특이적 프로모터, 세포형 특이적 프로모터, 등), 등.
일부 경우에서, 부위 지향적 변형 폴리펩타이드는 아미노산 서열을 포함하고, 이 서열은 도 3에서 묘사된 Cas9/Csn1 아미노산 서열의 아미노산 7-166 또는 731-1003과, 또는 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 상응하는 부분과 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 99%, 또는 100%, 아미노산 서열 동일성을 갖는다.
이식유전자 식물
상기에서 기재된 바와 같이, 일부 구현예에서, 당해 핵산 (예를 들면, 부위 지향적 변형 폴리펩타이드, 예를 들면, 자연 발생 Cas9; 변형된, 즉, 돌연변이된 또는 변이체, Cas9; 키메라 Cas9; 등를 인코딩하는 뉴클레오타이드 서열) 또는 당해 재조합 발현 벡터는 부위 지향적 변형 폴리펩타이드를 생산하는 이식유전자를 산출하기 위해 이식유전자로서 사용된다. 따라서, 본 발명은 추가로, 이식유전자 식물을 제공하고, 이 식물은 상기에서 기재된 바와 같이 당해 부위 지향적 변형 폴리펩타이드, 예를 들면, 자연 발생 Cas9; 변형된, 즉, 돌연변이된 또는 변이체, Cas9; 키메라 Cas9; 등를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산을 포함하는 이식유전자를 포함한다. 일부 구현예에서, 이식유전자 식물의 게놈은 당해 핵산을 포함한다. 일부 구현예에서, 이식유전자 식물은 유전적 변형에 대한 동종접합이다. 일부 구현예에서, 이식유전자 식물은 유전적 변형에 대한 이종접합이다.
외인성 핵산을 식물 세포에 도입하는 방법은 대상체기술에 공지되어 있다. 그와 같은 식물 세포는 상기에서 규정된 바와 같이 "형질전환된" 것으로 간주된다. 적당한 방법은 하기를 포함한다: 바이러스성 감염 (예컨대 이중가닥 DNA 바이러스), 형질감염, 콘주게이션, 원형질 융합, 전기천공, 입자총 기술, 칼슘 포스페이트 침전, 직접적인 미세주입, 실리콘 카바이드 위스커스(whiskers) 기술, 아그로박테리움-매개된 형질전환 등. 방법의 선택은 형질전환될 세포이 유형 형질전환이 일어나고 있는 상황 (즉 시험관내, 생체외, 또는 생체내)에 일반적으로 의존적이다.
토양 박테리아 아그로박테리움 투머파씨엔쓰를 기반으로 하는 형질전환 방법은 외인성 핵산 분자를 관다발 식물에 도입하는데 특히 유용하다. 야생형 형태의 아그로박테리움은 숙주 식물 상의 종양유발성 근두 암종 성장의 생산을 겨냥하는 Ti (종양-유도)를 함유한다. Ti 플라스미드의 종양-유도 T-DNA 영역의 식물 게놈으로의 이동은 Ti 플라스미드-인코딩된 발병력 유전자 뿐만 아니라 이동될 영역을 묘사하는 직접적인 DNA 반복체의 세트인 T-DNA 가장자리를 필요로 한다. 아그로박테리움-기반 벡터는 변형된 형태의 Ti 플라스미드이고, 여기서 종양 유도 기능은 식물 숙주에 도입될 관심 핵산 서열에 이해 대체된다.
아그로박테리움-매개된 형질전환은 일반적으로 상호삽입 벡터 또는 2원 벡터 시스템을 이용하고, 여기서 Ti 플라스미드의 성분은 아그로박테리움 숙주에서 영구적으로 남아 있고, 발병력 유전자를 운반하는 헬퍼 벡터와, T-DNA 서열에 의해 제한된 관심 유전자를 함유하는 셔틀 벡터 사이에서 분할된다. 다양한 2원 벡터는 대상체기술에서 잘 알려져 있고 예를 들면, Clontech (Palo Alto, Calif.)로부터 상업적으로 이용가능하다. 배양된 식물 세포 또는 상처난 조직 예컨대 잎 조직, 뿌리 외식편, 다자엽식물, 줄기 피스 또는 덩이줄기로 아그로박테리움을 공배양하는 방법은, 예를 들면, 또한 대상체기술에 공지되어 있다. 참고, 예를 들면, Glick 및 Thompson, (eds.), Methods in Plant Molecular Biology and Biotechnology, Boca Raton, Fla.: CRC Press (1993).
미세투사물 -매개된 형질전환은 또한 당해 이식유전자 식물을 생산하기 위해 사용될 수 있다. Klein 등 (Nature 327:70-73 (1987))에 의해 먼저 기재된 이러한 방법은, 염화칼슘, 스페르미딘 또는 폴리에틸렌 글리콜에 의한 침전에 의해 원하는 핵산 분자로 코팅된 미세투사물 예컨대 금 또는 텅스텐에 의존한다. 미세투사물 입자는 디바이스 예컨대 BIOLISTIC PD-1000 (Biorad; Hercules Calif.)를 사용하여 속씨고속 식물로 고속으로 가속화된다.
당해 핵산은, 핵산이, 예를 들면, 생체내 또는 생체외 프로토콜을 통해 식물 세포(들)에 들어갈 수 있는 방식으로 식물에 도입될 수 있다 "생체내"란, 예를 들면 침윤에 의해 살아있는 식물체에 투여된다는 것을 의미한다. "생체외"란, 세포 또는 외식편이 식물 밖에서 변형되고, 그 다음 그와 같은 세포 또는 기관이 식물로 재생되는 것을 의미한다. 하기에서 기재된 것을 포함하는,식물 세포의 안정한 형질전환에 또는 이식유전자 식물의 확립에 적당한 수많은 벡터가 기재되었다: Weissbach 및 Weissbach, (1989) Methods for Plant Molecular Biology Academic Press, and Gelvin 등, (1990) Plant Molecular Biology Manual, Kluwer Academic Publishers. 구체적인 예들은 아그로박테리움 투머파씨엔쓰의 Ti 플라스미드로부터 유도된 것들, 뿐만 아니라 하기에 의해 기재된 것들을 포함한다: Herrera-Estrella 등 (1983) Nature 303: 209, Bevan (1984) Nucl Acid Res. 12: 8711-8721, Klee (1985) Bio/Technolo 3: 637-642. 대안적으로, 비-Ti 벡터는 유리 DNA 전달 기술을 사용하여 DNA를 식물 및 세포로 이동시키기 위해 사용될 수 있다다. 이들 방법을 사용하여 이식유전자 식물 예컨대 밀, 쌀 (Christou (1991) Bio/Technology 9:957-9 및 4462) 및 옥수수 (Gordon-Kamm (1990) Plant Cell 2: 603-618)가 생산될 수 있다. 미성숙한 배아는 또한 하기에 대한 외떡잎식물의 우수한 표적 조직일 수 있다: 입자총 사용에 의한 직접적인 DNA 전달 기술 (Weeks 등 (1993) Plant Physiol 102: 1077-1084; Vasil (1993) Bio/Technolo 10: 667-674; Wan 및 Lemeaux (1994) Plant Physiol 104: 37-48 및 아그로박테리움-매개된 DNA 이동 (Ishida 등 (1996) Nature Biotech 14: 745-750). DNA를 엽록체에 도입하기 위한 예시적인 방법은 바이오리스틱 폭격, 원형질의 폴리에틸렌 글리콜 형질전환, 및 미세주입이다 (Danieli 등 Nat. Biotechnol 16:345-348, 1998; Staub 등 Nat. Biotechnol 18: 333-338, 2000; O'Neill 등 Plant J. 3:729-738, 1993; Knoblauch 등 Nat. Biotechnol 17: 906-909; 미국 특허 번호 5,451,513, 5,545,817, 5,545,818, 및 5,576,198; 국제출원 번호 WO 95/16783; 및 Boynton 등, Methods in Enzymology 217: 510-536 (1993), Svab 등, Proc. Natl. Acad. Sci. USA 90: 913-917 (1993), 및 McBride 등, Proc. Nati. Acad. Sci. USA 91: 7301-7305 (1994)). 바이오리스틱 폭격, 원형질의 폴리에틸렌 글리콜 형질전환 및 미세주입의 방법에 적당한 임의의 벡터는 엽록체 형질전환을 위한 표적화 벡터로서 적당한 것이다. 임의의 이중가닥 DNA 벡터는, 특히 도입의 방법이 아그로박테리움을 이용하지 않을 때 형질전환 벡터로서 사용될 수 있다.
유전적으로 변형될 수 있는 식물은 알갱이, 사료 작물, 과일, 체소류, 오일 씨드 농작물, 야자나무, 삼림, 및 포도나무를 포함한다. 변형될 수 있는 식물이 구체적인 예는 하기와 같다: 옥수수, 바나나, 땅콩, 완두콩, 해바라기, 토마토, 카놀라, 담배, 밀, 보리, 귀리, 감자, 대두, 면, 카네이션, 수수, 루핀 및 쌀.
형질전환된 식물 세포, 조직, 식물 및 형질전환된 식물 세포를 함유하는 생성물이 본 발명에 의해 또한 제공된다. 당해 형질전환된 세포, 및 조직 및 이들을 포함하는 생성물의 특징은 게놈에 통합된 당해 핵산의 존재, 및 부위 지향적 변형 폴리펩타이드, 예를 들면, 자연 발생 Cas9; 변형된, 즉, 돌연변이된 또는 변이체, Cas9; 키메라 Cas9; 등의 식물 세포에 의한 생산이다. 본 발명의 재조합 식물 세포는 재조합 세포의 모집단으로서, 또는 조직, 씨드, 전체의 식물, 줄기, 과일, 잎, 뿌리, 꽃, 줄기, 덩이줄기, 그레인, 동물 사료, 식물 분야, 등으로서 유용하다.
부위 지향적 변형 폴리펩타이드 (예를 들면, 자연 발생 Cas9; 변형된, 즉, 돌연변이된 또는 변이체, Cas9; 키메라 Cas9; 등)을 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산은 (예를 들면, 핵산이 무작위로 숙주세포 게놈에 통합될 때) 미공지된 프로모터의 조절하에 있을 수 있거나 (즉, 그것에 작동가능하게 연결될 수 있고), 공지된 프로모터의 조절 하에 있을 수 있다 (즉, 그것에 작동가능하게 연결될 수 있다). 적당한 공지된 프로모터는 임의의 공지된 프로모터일 수 있고 항시적으로 활성인 프로모터, 유도성 프로모터, 공간적으로 제한된 및/또는 일시적으로 제한된 프로모터, 등을 포함한다.
일부 경우에서, 부위 지향적 변형 폴리펩타이드는 아미노산 서열을 포함하고, 이 서열은 도 3에서 묘사된 Cas9/Csn1 아미노산 서열의 아미노산 7-166 또는 731-1003과, 또는 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 상응하는 부분과 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 99%, 또는 100%, 아미노산 서열 동일성을 갖는다.
당해 이식유전자 식물의 재생산적인 물질이 본 발명에 의해 또한 제공되고, 여기서 재생산적인 물질은 종자, 자손 식물 및 클론 물질을 포함한다.
정의- 파트 II
핵산, 폴리펩타이드, 세포 또는 유기체에 적용될 때 본원에서 사용된 용어 "자연-발생" 또는 "비변형된"은 자연에서 발견되는 핵산, 폴리펩타이드, 세포 또는 유기체를 의미한다. 예를 들면, 자연에서의 공급원으로부터 단리될 수 있고, 실험실에서 인간에 의해 의도적으로 변형되지 않은 유기체(바이러스 포함)에 존재하는 폴리펩타이드 또는 폴리뉴클레오타이드 서열은 자연 발생적이다.
본원에 사용된 "이종기원"은 각각 천연 핵산 또는 단백질에서 발견되지 않은 뉴클레오타이드 또는 폴리펩타이드 서열을 의미한다. 예를 들면, 융합 변이체 Cas9 부위 지향적 폴리펩타이드에서, 변이체 Cas9 부위 지향적 폴리펩타이드는 이종기원 폴리펩타이드(즉, Cas9 이외의 폴리펩타이드)에 융합될 수 있다. 이종기원 폴리펩타이드는 융합 변이체 Cas9 부위 지향적 폴리펩타이드에 의해 또한 나타낼 활성(예: 효소 활성)을 나타낼 수 있다. 이종기원 핵산 서열은 융합 변이체 Cas9 부위 지향적 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 생성하기 위해 변이체 Cas9 부위 지향적 폴리펩타이드(예: 유전공학에 의해)에 연결시킬 수 있다.
용어 "키메라 폴리펩타이드"는 자연 발생적이 아닌, 예를 들면, 인간 개입을 통해 아미노 서열의 두 개의 달리 분리된 세그먼트의 인공 조합으로 제조된 폴리펩타이드를 의미한다. 따라서, 키메라 폴리펩타이드는 또한 인간 개입의 결과이다. 따라서, 키메라 아미노산 서열을 포함하는 폴리펩타이드는 키메라 폴리펩타이드이다.
"부위 지향적 변형 폴리펩타이드" 또는 " RNA-결합 부위 지향적 폴리펩타이드" 또는 "RNA-결합 부위 지향적 폴리펩타이드"란 RNA를 결합하고, 특이적 DNA 서열에서 표적화되는 폴리펩타이드를 의미한다. 본원에서 기재된 바와 같은 부위 지향적 폴리펩타이드는 그것이 결합되는 RNA 분자에 의해 특이적 DNA 서열에서 표적화된다. RNA 분자는 표적 DNA 내에서 표적 서열에 상보적인 서열을 포함하고, 따라서 표적 DNA(표적 서열) 내의 특이적 위치에서 결합된 폴리펩타이드를 표적화한다.
일부 구현예에서, 당해 핵산(예: DNA-표적화 RNA, DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산; 부위 지향적 폴리펩타이드를 인코딩하는 핵산 등)은 추가의 바람직한 특성(예: 변형되거나 조절된 안정성, 준세포 표적화; 추적, 예를 들면, 형광 라벨; 단백질 또는 단백질 복합체용 결합 부위 등)을 제공하는 변형 또는 서열을 포함한다. 비제한적인 예는 5' 캡(예: 7-메틸구아닐레이트 캡(m7G)); 3' 폴리아데닐화 꼬리(즉, 3' 폴리(A) 꼬리); 리보스위치 서열(예: 단백질 및/또는 단백질 복합체에 의한 조절된 안정성 및/또는 조절된 접근성을 허용하는); RNA를 준세포 위치(예: 핵, 미토콘드리아, 엽록체 등)에서 표적화하는 변형 또는 서열; 추적(예: 형광성 분자에 직접 접합, 형광 검출을 촉진시키는 잔기에 접합, 형광 검출을 허용하는 서열 등)을 제공하는 변형 또는 서열; 단백질(예: 전사 활성제, 전사 억제인자. DNA 메틸트랜스퍼라제, DNA 데메틸라제, 히스톤 아세틸트랜스퍼라제, 히스톤 데아세틸라제 등을 포함하는 DNA에 작용하는 단백질)에 결합 부위를 제공하는 변형 또는 서열; 및 이들의 조합을 포함한다.
일부 구현예에서, DNA-표적화 RNA는 상기 기재된 임의의 특징을 제공하는 5' 또는 3' 말단에 추가의 세그먼트를 포함한다. 예를 들면, 적합한 제 3 세그먼트는 5' 캡(예: 7-메틸구아닐레이트 캡(m7G)); 3' 폴리아데닐화 꼬리(즉, 3' 폴리(A) 꼬리); 리보스위치 서열(예: 단백질 및/또는 단백질 복합체에 의한 조절된 안정성 및/또는 조절된 접근성을 허용하는); RNA를 준세포 위치(예: 핵, 미토콘드리아, 엽록체 등)에서 표적화하는 서열; 추적(예: 형광성 분자에 직접 접합, 형광 검출을 촉진시키는 잔기에 접합, 형광 검출을 허용하는 서열 등)을 제공하는 변형 또는 서열; 단백질(예: 전사 활성제, 전사 억제인자. DNA 메틸트랜스퍼라제, DNA 데메틸라제, 히스톤 아세틸트랜스퍼라제, 히스톤 데아세틸라제 등을 포함하는 DNA에 작용하는 단백질)에 결합 부위를 제공하는 변형 또는 서열; 및 이들의 조합을 포함할 수 있다.
당해 DNA-표적화 RNA 및 당해 부위 지향적 폴리펩타이드는 복합체를 형성한다(즉, 비공유 상호작용을 통해 결합한다). DNA-표적화 RNA는 표적 DNA의 서열에 상보적인 뉴클레오타이드 서열을 포함함으로써 복합체에 표적 특이성을 제공한다. 복합체의 부위 지향적 폴리펩타이드는 부위-특이적 활성을 제공한다. 환언하면, 부위 지향적 폴리펩타이드는 DNA-표적화 RNA의 단백질-결합 세그먼트과의 회합에 의해 표적 DNA 서열(예: 염색체 핵산 중의 표적 서열; 염색체외 핵산, 예를 들면, 에피솜 핵산, 미니써클 등 중의 표적 서열; 미토콘드리아 핵산 중의 표적 서열; 엽록체 핵산 중의 표적 서열; 플라스미드 중의 표적 서열 등)에 유도된다.
일부 구현예에서, 당해 DNA-표적화 RNA는 두 개의 별도의 RNA 분자(RNA 폴리뉴클레오타이드)를 포함하고, 본원에서 "이중-분자 DNA-표적화 RNA" 또는 "두-분자 DNA-표적화 RNA"로서 칭명된다. 기타 구현예에서, 당해 DNA-표적화 RNA는 단일 RNA 분자(단일 RNA 폴리뉴클레오타이드)이고, 본원에서 "단일-분자 DNA-표적화 RNA"로서 칭명된다. 달리 구체화되지 않는 한, 용어 "DNA-표적화 RNA"는 포괄적이어서, 단일-분자 DNA-표적화 RNA 및 이중-분자 DNA-표적화 RNA 둘 다를 언급한다.
당해 두-분자 DNA-표적화 RNA는 두 개의 별도의 RNA 분자("타겟터-RNA" 및 "활성제-RNA")를 포함한다. 당해 두-분자 DNA-표적화 RNA의 두 RNA 분자 각각은 두 RNA 분자의 상보적 뉴클레오타이드가 하이브리드화되어 단백질 결합 세그먼트의 이중 가닥 RNA 듀플렉스를 형성하도록 서로 상보적인 뉴클레오타이드의 스트레치를 포함한다.
당해 단일-분자 DNA-표적화 RNA는 서로 상보적인 뉴클레오타이드의 스트레치(타겟터-RNA 및 활성제-RNA)를 포함하고, 개재 뉴클레오타이드("링커" 또는 "링커 뉴클레오타이드")에 의해 공유 결합되고, 하이브리드화하여 단백질-결합 세그먼트의 이중 가닥 RNA 듀플렉스(dsRNA 듀플렉스)를 형성하고, 따라서 줄기-루프 구조를 유도한다. 타겟터-RNA 및 활성제-RNA는 타겟터-RNA의 3' 말단 및 활성제-RNA의 5' 말단을 통해 공유 결합될 수 있다. 대안적으로, 타겟터-RNA 및 활성제-RNA는 타겟터-RNA의 5' 말단 및 활성제-RNA의 3' 말단을 통해 공유 결합될 수 있다.
예시적인 두-분자 DNA-표적화 RNA는 crRNA-형("CRISPR RNA" 또는 "타겟터-RNA" 또는 "crRNA" 또는 "crRNA 반복체") 분자 및 상응하는 tracrRNA-형("트랜스-작용성 CRISPR RNA" 또는 "활성제-RNA" 또는 "tracrRNA") 분자를 포함한다. crRNA-형 분자(타겟터-RNA)는 DNA-표적화 RNA의 DNA-표적화 세그먼트(단일 가닥) 및 DNA-표적화 RNA의 단백질-결합 세그먼트의 dsRNA 듀플렉스의 1/2을 형성하는 뉴클레오타이드의 스트레치("듀플렉스 형성 세그먼트") 모두를 포함한다. 상응하는 tracrRNA-형 분자(활성제-RNA)는 DNA-표적화 RNA의 단백질-결합 세그먼트의 dsRNA 듀플렉스의 나머지 1/2을 형성하는 뉴클레오타이드의 스트레치(듀플렉스 형성 세그먼트)를 포함한다. 환언하면, crRNA-형 분자의 뉴클레오타이드의 스트레치는 tracrRNA-형 분자의 뉴클레오타이드의 스트레치에 상보적이고, 이와 하이브리드화하여 DNA-표적화 RNA의 단백질-결합 도메인의 dsRNA 듀플렉스를 형성한다. 이와 같이, 각각의 crRNA-형 분자는 상응하는 tracrRNA-형 분자를 갖는다고 할 수 있다. crRNA-형 분자는 추가로 단일 가닥 DNA-표적화 세그먼트를 제공한다. 따라서, crRNA-형 및 tracrRNA-형 분자(상응하는 쌍으로서)가 하이브리드화하여 DNA-표적화 RNA를 형성한다. 주어진 crRNA 또는 tracrRNA 분자의 정확한 서열은 RNA 분자가 발견되는 종의 특징이다.
용어 "활성제-RNA"는 이중-분자 DNA-표적화 RNA의 tracrRNA-형 분자를 의미하기 위해 본원에서 사용된다. 용어 "타겟터-RNA"는 이중-분자 DNA-표적화 RNA의 crRNA-형 분자를 의미하기 위해 본원에서 사용된다. 용어 "듀플렉스-형성 세그먼트"은 상응하는 활성제-RNA 또는 타겟터-RNA 분자의 뉴클레오타이드의 스트레치로 하이브리드화함으로써 dsRNA 듀플렉스의 형성에 기여하는 활성제-RNA 또는 타겟터-RNA의 뉴클레오타이드의 스트레치를 의미하기 위해 본원에서 사용된다. 환언하면, 활성제-RNA는 상응하는 타겟터-RNA의 듀플렉스-형성 세그먼트에 상보적인 듀플렉스-형성 세그먼트를 포함한다. 이와 같이, 활성제-RNA는 듀플렉스-형성 세그먼트를 포함하는 반면, 타겟터-RNA는 듀플렉스-형성 세그먼트 및 DNA-표적화 RNA의 DNA-표적화 세그먼트 둘 다를 포함한다. 따라서, 당해 이중-분자 DNA-표적화 RNA는 임의의 상응하는 활성제-RNA 및 타겟터-RNA 쌍으로 이루어질 수 있다.
두-분자 DNA-표적화 RNA는 타겟터-RNA와 활성제-RNA의 조절된(즉, 조건부) 결합을 허용하도록 설계될 수 있다. 활성제-RNA 및 타겟터-RNA 모두가 dCas9를 갖는 기능성 복합체에 결합되지 않는 한 두-분자 DNA-표적화 RNA가 기능성이 아니기 때문에, 두-분자 DNA-표적화 RNA는 활성제-RNA 및 타겟터-RNA 사이의 결합을 유도성이도록 함으로써 유도성(예: 약물 유도성)일 수 있다. 하나의 비제한적인 예로서, RNA 압타머를 사용하여 활성제-RNA와 타겟터-RNA의 결합을 조절(억제)할 수 있다. 따라서, 활성제-RNA 및/또는 타겟터-RNA는 RNA 압타머 서열을 포함할 수 있다.
RNA 압타머는 당해 기술 분야에 공지되어 있고, 일반적으로 리보스위치의 합성 버전이다. 용어들 "RNA 압타머" 및 "리보스위치"는 그들이 일부인 RNA 분자의 구조의 유도성 조절 (및 따라서 특이적 서열의 이용가능성)을 제공하는 합성 및 천연 핵산 서열 둘 다를 포함하기 위해 본원에서 상호교환적으로 사용된다. RNA 압타머는 보통 특이적으로 특정 약물(예: 소분자)을 결합하는 특정 구조(예: 헤어핀)에 폴딩되는 서열을 포함한다. 약물의 결합은 RNA의 폴딩에서 구조적 변화를 일으키고, 이는 압타머가 일부인 핵산의 특징을 변화시킨다. 비제한적인 예로서: (i) 압타머를 갖는 활성제-RNA는, 압타머가 적절한 약물로 결합되지 않는 한 동족 타겟터-RNA에 결합할 수 없을 수 있고; (ii) 압타머를 갖는 타겟터-RNA는, 압타머가 적절한 약물로 결합되지 않는 한 동족 활성제-RNA에 결합할 수 없을 수 있고; (iii) 각각 상이한 약물을 결합하는 상이한 압타머를 포함하는 타겟터-RNA 및 활성제-RNA는 두 약물이 존재하지 않은 한, 서로 결합할 수 없을 수 있다. 이러한 예에 의해 실증된 바와 같이, 두-분자 DNA-표적화 RNA는 유도성이도록 설계될 수 있다.
압타머 및 리보스위치의 예는, 예를 들면, 전문이 본원에 참조로 인용된 문헌(참조: Nakamura 등, Genes Cells. 2012 May;17(5):344-64; Vavalle 등, Future Cardiol. 2012 May;8(3):371-82; Citartan 등, Biosens Bioelectron. 2012 Apr 15;34(1):1-11; 및 Liberman 등, Wiley Interdiscip Rev RNA. 2012 May-Jun;3(3):369-84)에서 찾을 수 있다.
두-분자 DNA-표적화 RNA에 포함될 수 있는 뉴클레오타이드 서열의 비제한적인 예는 서열번호 671-678에 나타낸 임의의 하나의 활성제 RNA 세트이 듀플렉스 형성 세그먼트과 쌍을 이룰 수 있는 타겟터 RNA(예: 서열번호 566-567)를 포함한다.
예시적인 단일-분자 DNA-표적화 RNA는 하이브리드화하여 dsRNA 듀플렉스를 형성하는 뉴클레오타이드의 두 개의 상보적 스트레치를 포함한다. 일부 구현예에서, 단일-분자 DNA-표적화 RNA(또는 스트레치를 인코딩하는 DNA)의 뉴클레오타이드의 두 상보적 스트레치 중의 하나는 적어도 8개 인접 뉴클레오타이드의 스트레치에 대해 서열번호 431-562에 나타낸 활성제-RNA(tracrRNA) 서열 중의 하나와 적어도 약 60% 동일하다. 예를 들면, 단일-분자 DNA-표적화 RNA(또는 스트레치를 인코딩하는 DNA)의 뉴클레오타이드의 두 상보적 스트레치 중의 하나는 적어도 8개 인접 뉴클레오타이드의 스트레치에 대해 서열번호 431-562에 나타낸 tracrRNA 서열 중의 하나와 적어도 약 65% 동일, 적어도 약 70% 동일, 적어도 약 75% 동일, 적어도 약 80% 동일, 적어도 약 85% 동일, 적어도 약 90% 동일, 적어도 약 95% 동일, 적어도 약 98% 동일, 적어도 약 99% 동일 또는 100% 동일하다.
일부 구현예에서, 단일-분자 DNA-표적화 RNA(또는 스트레치를 인코딩하는 DNA)의 뉴클레오타이드의 두 상보적 스트레치 중의 하나는 적어도 8개 인접 뉴클레오타이드의 스트레치에 대해 서열번호 563-679에 나타낸 타겟터-RNA(crRNA) 서열 중의 하나와 적어도 약 60% 동일하다. 예를 들면, 단일-분자 DNA-표적화 RNA(또는 스트레치를 인코딩하는 DNA)의 뉴클레오타이드의 두 상보적 스트레치 중의 하나는 적어도 8개 인접 뉴클레오타이드의 스트레치에 대해 서열번호 563-679에 나타낸 crRNA 서열 중의 하나와 적어도 약 65% 동일, 적어도 약 70% 동일, 적어도 약 75% 동일, 적어도 약 80% 동일, 적어도 약 85% 동일, 적어도 약 90% 동일, 적어도 약 95% 동일, 적어도 약 98% 동일, 적어도 약 99% 동일 또는 100% 동일하다.
상기에서와 같이, 본원에 사용된 "숙주 세포"는 생체내 또는 시험관내 진핵 세포, 원핵 세포(예: 박테리아 또는 고세균 세포), 또는 단세포 독립체로서 배양된 다중세포 유기체(예: 세포주)로부터의 세포를 나타내고, 이 진핵 또는 원핵 세포가 핵산을 위한 수령체로서 사용될 수 있거나 사용되었고, 핵산에 의해 형질전환된 본래 세포의 자손을 포함한다. 단일 세포의 자손은 천연, 우발적 또는 계획적 돌연변이에 기인하여, 원래 부모와 형태 또는 게놈 또는 총 DNA 보체가 반드시 완전히 동일할 필요가 없을 수 있는 것으로 이해된다. "재조합 숙주 세포"(또한 "유전적으로 변형된 숙주 세포"로서 칭명됨)는 이종기원 핵산, 예를 들면, 발현 벡터가 도입된 숙주 세포이다. 예를 들면, 당해 박테리아 숙주 세포는 외인성 핵산(예: 플라스미드 또는 재조합 발현 벡터)의 적당한 박테리아 숙주 세포에 도입함으로써 유전적으로 변형된 박테리아 숙주 세포이고, 당해 진핵 숙주 세포는 외인성 핵산의 적당한 진핵 숙주 세포에 도입시킴으로써 유전적으로 변형된 진핵 숙주 세포(예: 포유동물 생식 세포)이다.
"정의- 파트 I"에 제공된 정의는 또한 본 섹션에도 적용가능하고; 용어의 추가의 설명을 위해 "정의- 파트 I"을 참조한다.
본 발명이 추가로 기재되기 전에, 본 발명은 물론 변할 수 있기 때문에, 기재된 특정 구현예에 국한되지 않는 것으로 이해되어야 한다. 또한, 본원에 사용된 용어는 단지 특정 구현예를 기술하기 위한 것이고, 본 발명의 범위가 첨부된 특허청구범위에 의해서만 제한되기 때문에, 제한하고자 하지 않는다고 이해되어야 한다.
값의 범위가 제공될 경우, 각각의 개재 값, 문맥이 명백하게 달리 지시하지 않는 한 그 범위의 하한치의 단위의 1/10까지, 그 범위의 상한치와 하한치 사이 및 그 기술된 범위 내의 임의의 다른 언급된 또는 개입 값이 본 발명 내에 포함된다고 이해된다. 이들 더 작은 범위의 상한치 및 하한치는 독립적으로 더 작은 범위에 포함될 수 있고, 또한 언급된 범위에서 임의의 특이적으로 제외된 한계까지 본 발명 대상체내에 포함된다. 언급된 범위가 한계치 중의 하나 또는 둘 다를 포함할 경우, 포함된 한계치 중의 하나 또는 둘 다를 제외하는 범위도 또한 본 발명에 포함된다.
달리 정의되지 않으면, 본원에 사용된 모든 기술 및 과학 용어들은 본 발명이 속하는 당해 분야의 숙련가에 의해 전형적으로 이해되는 바와 동일한 의미를 갖는다. 본원에서 기술된 것들과 유사하거나 등가인 임의의 방법 및 물질이 또한 본 발명의 실시 또는 시험에 사용될 수 있지만, 바람직한 방법 및 물질은 이하 기술된다. 본원에서 언급된 모든 공보들은 인용된 공보와 관련하여 방법 및/또는 물질을 나타내고 기술하기 위해 본원에 참조로 인용된다.
본원 및 첨부된 특허청구범위에서 사용된 바와 같이, 단수 형태 "a", "an" 및 "the"는 문맥이 명확히 달리 지시하지 않는 한 복수 지시대상을 포함한다는 것을 주시해야 한다. 따라서, 예를 들면, "효소적으로 불활성 Cas9 폴리펩타이드"에 대한 참조는 이러한 폴리펩타이드의 복수를 포함하고, "표적 핵산"에 대한 참조는 하나 이상의 표적 핵산 및 당해 기술 분야의 숙련가에게 공지된 이의 등가물 등에 대한 참조를 포함한다. 또한, 특허청구범위가 임의의 선택적 요소를 제외하도록 드래프트될 수 있다는 것이 주시된다. 이와 같이, 이 서술은 청구항 요소의 설명 또는 "음성" 한계의 사용과 관련하여 "유일하게", "단지" 등과 같은 이러한 배타적인 용어의 사용에 대한 선행된 기준으로서 작용하도록 의도된다.
명쾌함을 위해, 개별적 구현예의 문맥에서 기재된 본 발명의 특정의 특징은 또한 단일 구현예에 조합으로 제공될 수 있다고 인정된다. 반대로, 간결함을 위해 단일 구현예의 문맥에서 기재된 본 발명의 다양한 특징은 또한 별도로 또는 임의의 적합한 아-조합으로 제공될 수 있다. 본 발명에 속하는 구현예의 모든 조합은 구체적으로 본 발명에 의해 포함되고, 마치 각각 및 모든 조합이 개별적으로 명백하게 개시된 것처럼 본원에서 개시된다. 또한, 다양한 구현예 및 이의 요소의 모든 아-조합은 또한 본 발명에 의해 구체적으로 포함되고, 마치 각각 및 모든 이러한 아-조합이 개별적으로 명백하게 본원에서 개시된 것처럼 본원에서 개시된다.
본원에서 논의된 공보들은 단지 본원의 출원일 이전의 이들의 개시내용을 위해 제공된다. 본원에서 어떤 것도 본 발명이 선행 발명에 의해 이러한 공보를 선행할 자격이 없음을 승인하는 것으로 해석되어서는 안된다. 게다가, 제공된 공고일은 독립적으로 확인될 필요가 있을 수 있는 실제 공고일과 상이할 수 있다.
상세한 설명- 파트 II
본 명세서는 숙주 세포에서 표적 핵산의 전사를 조절하는 방법을 제공한다. 상기 방법은 일반적으로 표적 핵산을 효소적 불활성 Cas9 폴리펩타이드 및 단일-가이드 RNA와 접촉시킴을 포함한다. 상기 방법은 다양한 적용에 유용하고, 이는 또한 제공된다.
본 명세서의 전사 조절 방법은 RNAi를 포함하는 방법의 결점 중의 일부를 극복한다. 본 명세서의 전사 조절 방법은 연구 적용, 약물 발견(예: 고처리량 스크리닝), 표적 검증, 산업적 적용(예: 작물 공학, 미생물 공학 등), 진단 적용, 치료적 적용 및 이미지화 기술을 포함하는 다양한 적용에서 용도를 발견한다.
전사 조절 방법
본 명세서는 숙주 세포에서 표적 DNA의 전사를 선택적으로 조절하는 방법을 제공한다. 상기 방법은 일반적으로 a) 숙주 세포에 i) DNA-표적화 RNA, 또는 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산, 및 ii) 변이체 Cas9 부위 지향적 폴리펩타이드("변이체 Cas9 폴리펩타이드") 또는 변이체 Cas9 폴리펩타이드를 인코딩하는 뉴클레오타이드를 포함하는 핵산을 도입함을 포함하고, 여기서 변이체 Cas9 폴리펩타이드는 감소된 엔도데옥시리보뉴클레아제 활성을 나타낸다.
DNA-표적화 RNA(또한 본원에서 "crRNA" 또는 "가이드 RNA" 또는 "gRNA"으로서 칭명된다)는 i) 표적 DNA 중의 표적 서열에 상보적인 뉴클레오타이드 서열을 포함하는 제 1 세그먼트; ii) 부위 지향적 폴리펩타이드와 상호작용하는 제 2 세그먼트; 및 iii) 전사 종결자를 포함한다. 표적 DNA 중의 표적 서열에 상보적인 뉴클레오타이드 서열을 포함하는 제 1 세그먼트는 본원에서 "표적화 세그먼트"로서 칭명된다. 부위 지향적 폴리펩타이드와 상호작용하는 제 2 세그먼트는 또한 본원에서 "단백질 결합 서열" 또는 "dCas9-결합 헤어핀" 또는 "dCas9 핸들"로서 칭명된다. "세그먼트"란, 분자의 세그먼트/섹션/영역, 예를 들어, RNA 중의 뉴클레오타이드의 인접 스트레치를 의미한다. "세그먼트"의 정의는 특별한 문맥에서 달리 특이적으로 정의되지 않는 한, 특정 수의 총 염기쌍에 국한되지 않고, 임의의 총 길이인 RNA 분자의 영역을 포함할 수 있고, 다른 분자에 상보성을 갖는 영역을 포함할 수 있거나 포함하지 않을 수 있다. 본 명세서에 따르는 DNA-표적화 RNA는 단일 RNA 분자(단일 RNA 폴리뉴클레오타이드)일 수 있고, 이는 본원에서 "단일-분자 DNA-표적화 분자", "단일-가이드 RNA" 또는 "sgRNA"로서 칭명될 수 있다. 본 명세서에 따르는 DNA-표적화 RNA는 2개의 RNA 분자를 포함할 수 있다. 용어 "DNA-표적화 RNA" 또는 "gRNA"는 포괄적이어서, 두-분자 DNA-표적화 RNA 및 단일-분자 DNA-표적화 RNA(즉, sgRNA) 둘 다를 언급한다.
변이체 Cas9 부위 지향적 폴리펩타이드는 i) DNA-표적화 RNA와 상호작용하는 RNA-결합 부분; 및 ii) 감소된 엔도데옥시리보뉴클레아제 활성을 나타내는 활성 부분을 포함한다.
DNA-표적화 RNA 및 변이체 Cas9 폴리펩타이드는 숙주 세포에서 복합체를 형성하고, 상기 복합체는 숙주 세포에서 표적 DNA의 전사를 선택적으로 조절한다.
일부 경우에서, 본 명세서의 전사 조절 방법은 숙주 세포에서 표적 핵산의 선택적 조절(예: 감소 또는 증가)을 제공한다. 예를 들면, 표적 핵산의 전사의 "선택적 감소"는 표적 핵산의 전사를 DNA-표적화 RNA/변이체 Cas9 폴리펩타이드 복합체의 부재시의 표적 핵산의 전사 수준과 비교하여 적어도 약 10%, 적어도 약 20%, 적어도 약 30%, 적어도 약 40%, 적어도 약 50%, 적어도 약 60%, 적어도 약 70%, 적어도 약 80%, 적어도 약 90%, 또는 90%보다 더 크게 감소시킨다. 표적 핵산의 전사의 선택적 감소는 표적 핵산의 전사를 감소시키지만, 실질적으로 비-표적 핵산의 전사는 감소시키지 않는다, 예를 들면, 비-표적 핵산의 전사는 모든 경우 DNA-표적화 RNA/변이체 Cas9 폴리펩타이드 복합체의 부재시의 비-표적 핵산의 전사 수준과 비교하여 10% 미만으로 감소된다.
증가된 전사
표적 DNA의 "선택된" 증가된 전사는 표적 DNA의 전사를 DNA-표적화 RNA/변이체 Cas9 폴리펩타이드 복합체의 부재시의 표적 DNA의 전사 수준과 비교하여 적어도 약 1.1배(예: 적어도 약 1.2배, 적어도 약 1.3배, 적어도 약 1.4배, 적어도 약 1.5배, 적어도 약 1.6배, 적어도 약 1.7배, 적어도 약 1.8배, 적어도 약 1.9배, 적어도 약 2배, 적어도 약 2.5배, 적어도 약 3배, 적어도 약 3.5배, 적어도 약 4배, 적어도 약 4.5배, 적어도 약 5배, 적어도 약 6배, 적어도 약 7배, 적어도 약 8배, 적어도 약 9배, 적어도 약 10배, 적어도 약 12배, 적어도 약 15배 또는 적어도 약 20배) 증가시킬 수 있다. 표적 DNA의 전사의 선택적 증가는 표적 DNA의 전사를 증가시키지만, 비-표적 DNA의 전사는 실질적으로 증가시키지 않는다, 예를 들면, 비-표적 DNA의 전사는 모든 경우에 DNA-표적화 RNA/변이체 Cas9 폴리펩타이드 복합체의 부재시의 비-표적화된 DNA의 전사 수준과 비교하여 약 5배 미만(예: 약 4배 미만, 약 3배 미만, 약 2배 미만, 약 1.8배 미만, 약 1.6배 미만, 약 1.4배 미만, 약 1.2배 미만 또는 약 1.1배 미만) 증가된다.
비제한적인 예로서, 증가는 dCas9를 이종기원 서열에 융합시킴으로써 달성될 수 있다. 적합한 융합 파트너는 표적 DNA 또는 표적 DNA와 관련된 폴리펩타이드(예: 히스톤 또는 기타 DNA-결합 단백질) 상에서 직접 작용함으로써 전사를 간접적으로 증가시키는 활성을 제공하는 폴리펩타이드를 포함하지만, 이에 국한되지 않는다. 적합한 융합 파트너는 메틸트랜스퍼라제 활성, 데메틸라제 활성, 아세틸트랜스퍼라제 활성, 데아세틸라제 활성, 키나제 활성, 포스파타제 활성, 우비퀴틴 리가제 활성, 탈우비퀴틴화 활성, 아데닐화 활성, 탈아데닐화 활성, SUMO일화 활성, 탈SUMO일화 활성, 리보실화 활성, 탈리보실화 활성, 미리스토일화 활성 또는 탈미리스토일화 활성을 제공하는 폴리펩타이드를 포함하지만, 이에 국한되지 않는다.
추가의 적합한 융합 파트너는 표적 핵산의 증가된 전사를 직접 제공하는 폴리펩타이드(예: 전사 활성제 또는 이의 단편, 전사 활성제, 소분자/약물 반응성 전사 조절제를 모집하는 단백질 또는 이의 단편 등)를 포함하지만, 이에 국한되지 않는다.
원핵생물에서 전사를 증가시키기 위해 dCas9 융합 단백질을 사용하는 당해 방법의 비제한적인 예는 박테리아 1-하이브리드(B1H) 또는 2-하이브리드(B2H) 시스템의 변형을 포함한다. B1H 시스템에서, DNA 결합 도메인(BD)은 박테리아 전사 활성화 도메인(AD, 예를 들면, 에스케리치아 콜리 RNA 폴리머라제의 알파 서브유닛(RNAPα))에 융합된다. 따라서, 당해 dCas9는 AD를 포함하는 이종기원 서열에 융합될 수 있다. 당해 dCas9 융합 단백질이 프로모터(DNA-표적화 RNA에 의해 거기서 표적화된)의 업스트림 영역에 도달하면, dCas9 융합 단백질의 AD(예: RNAPα)는 RNAP 홀로효소를 모집하여 전사 활성화를 유도한다. B2H 시스템에서, BD는 AD에 직접 융합되지 않고; 대신에 이들의 상호작용은 단백질-단백질 상호작용(예: GAL11P-GAL4 상호작용)에 의해 매개된다. 당해 방법에 사용하기 위해 이러한 시스템을 변형시키기 위해, dCas9는 단백질-단백질 상호작용을 제공하는 제 1 단백질 서열(예: 효모 GAL11P 및/또는 GAL4 단백질)에 융합될 수 있고, RNAα는 단백질-단백질 상호작용을 완성하는 제 2 단백질 서열(예: GAL11P가 dCas9에 융합될 경우 GAL4, GAL4가 dCas9에 융합될 경우 GAL11P 등)에 융합될 수 있다. GAL11P와 GAL4 사이의 결합 친화도는 결합 및 전사 발사 속도의 효율을 증가시킨다.
진핵생물에서 전사를 증가시키기 위해 dCas9 융합 단백질을 사용하는 당해 방법의 비제한적인 예는 dCas9의 활성화 도메인(AD)(예: GAL4, 헤르페스바이러스 활성화 단백질 VP16 또는 VP64, 인간 핵 인자 NF-kB p65 서브유닛 등)에의 융합을 포함한다. 시스템을 유도성이도록 하기 위해, dCas9 융합 단백질의 발현은 유도성 프로모터(예: Tet-ON, Tet-OFF 등)에 의해 조절될 수 있다. DNA-표적화 RNA는 공지된 전사 반응 요소(예: 프로모터, 인핸서 등), 공지된 업스트림 활성화 서열(UAS), 표적 DNA의 발현을 제어할 수 있는 것으로 의심받는 공지되지 않거나 공지된 기능의 서열을 표적화하도록 설계할 수 있다.
추가의 융합 파트너
증가되거나 감소된 전사를 달성하기 위한 융합 파트너의 비제한적인 예는 도 54에 열거되고, 전사 활성제 및 전사 억제인자 도메인(예: 크뤼펠 관련 박스(KRAB 또는 SKD); Mad mSIN3 상호작용 도메인(SID); ERF 억제인자 도메인(ERD) 등)을 포함한다. 일부 이러한 경우에서, dCas9 융합 단백질은 DNA-표적화 RNA에 의해 표적 DNA 중의 특이적 위치(즉, 서열)에 표적화되고, 유전자좌-특이적 조절, 예를 들면, (선택적으로 전사 활성제 기능을 억제하는) 프로모터에 대한 RNA 폴리머라제 결합을 차단하고/하거나 국소 염색질 상태를 변형시킴(예: 표적 DNA를 변형시키거나 표적 DNA와 관련된 폴리펩타이드를 변형시키는 융합 서열이 사용될 경우)을 발휘한다. 일부 경우에서, 변화는 일시적이다(예: 전사 억제 또는 활성화). 일부 경우에서, 변화는 유전성이다(예: 후성유전적 변형이 표적 DNA 또는 표적 DNA와 관련되는 단백질, 예를 들면, 뉴클레오솜 히스톤에 수행되는 경우).
일부 구현예에서, 이종기원 서열은 dCas9 폴리펩타이드의 C-말단에 융합될 수 있다. 일부 구현예에서, 이종기원 서열은 dCas9 폴리펩타이드의 N-말단에 융합될 수 있다. 일부 구현예에서, 이종기원 서열은 dCas9 폴리펩타이드의 내부 부분(즉, N- 또는 C-말단 이외의 부분)에 융합될 수 있다.
당해 dCas9 융합 단백질을 사용하는 방법의 생물학적 효과는 임의의 편리한 방법(예: 유전자 발현 검정; 염색질-기반 검정, 예를 들면, 염색질 면역침전(ChiP), 염색질 생체내 검정(CiA) 등)에 의해 검출될 수 있다.
일부 경우에서, 당해 방법은 둘 이상의 상이한 DNA-표적화 RNA의 사용을 포함한다. 예를 들면, 2개의 상이한 DNA-표적화 RNA가 단일 숙주 세포에서 사용될 수 있고, 여기서, 2개의 상이한 DNA-표적화 RNA가 동일한 표적 핵산에서 2개의 상이한 표적 서열을 표적화한다.
따라서, 예를 들면, 당해 전사 조절 방법은 숙주 세포에 제 2 DNA-표적화 RNA, 또는 제 2 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산을 도입함을 추가로 포함할 수 있고, 여기서 제 2 DNA-표적화 RNA는 i) 표적 DNA 중의 제 2 표적 서열에 상보적인 뉴클레오타이드 서열을 포함하는 제 1 세그먼트; ii) 부위 지향적 폴리펩타이드와 상호작용하는 제 2 세그먼트; 및 iii) 전사 종결자를 포함한다. 일부 경우에서, 동일한 표적 핵산에서 2개의 상이한 표적화 서열을 표적화하는 2개의 상이한 DNA-표적화 RNA의 사용은 표적 핵산의 전사에서 증가된 조절(예: 감소 또는 증가)을 제공한다.
또 하나의 예로서, 2개의 상이한 DNA-표적화 RNA가 단일 숙주 세포에서 사용될 수 있고, 여기서 2개의 상이한 DNA-표적화 RNA는 두 개의 상이한 표적 핵산을 표적화한다. 따라서, 예를 들면, 당해 전사 조절 방법은 숙주 세포에 제 2 DNA-표적화 RNA 또는 제 2 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산을 도입함을 추가로 포함할 수 있고, 여기서 제 2 DNA-표적화 RNA는 i) 적어도 제 2 표적 DNA 중의 표적 서열에 상보적인 뉴클레오타이드 서열을 포함하는 제 1 세그먼트; ii) 부위 지향적 폴리펩타이드와 상호작용하는 제 2 세그먼트; 및 iii) 전사 종결자를 포함한다.
일부 구현예에서, 당해 핵산(예: DNA-표적화 RNA, 예를 들면, 단일-분자 DNA-표적화 RNA, 활성제-RNA, 타겟터-RNA 등; 공여체 폴리뉴클레오타이드; 부위 지향적 변형 폴리펩타이드를 인코딩하는 핵산 등)은 추가의 바람직한 특징(예: 변형되거나 조절된 안정성; 준세포 표적화; 추적, 예를 들면, 형광 라벨; 단백질 또는 단백질 복합체용 결합 부위 등)을 제공하는 변형 또는 서열을 포함한다. 비제한적인 예들은 5' 캡(예: 7-메틸구아닐레이트 캡(m7G)); 3' 폴리아데닐화 꼬리(즉, 3' 폴리(A) 꼬리); 리보스위치 서열 또는 압타머 서열(예: 단백질 및/또는 단백질 복합체에 의한 조절된 안정성 및/또는 조절된 접근성을 허용하는); 종결자 서열; dsRNA 듀플렉스(즉, 헤어핀)를 형성하는 서열; RNA를 준세포 위치(예: 핵, 미토콘드리아, 엽록체 등)에서 표적화하는 변형 또는 서열; 추적(예: 형광성 분자에 직접 접합, 형광 검출을 촉진시키는 잔기에 접합, 형광 검출을 허용하는 서열 등)을 제공하는 변형 또는 서열; 단백질(예: 전사 활성제, 전사 억제인자, DNA 메틸트랜스퍼라제, DNA 데메틸라제, 히스톤 아세틸트랜스퍼라제, 히스톤 데아세틸라제 등을 포함하는 DNA에 작용하는 단백질)에 결합 부위를 제공하는 변형 또는 서열; 및 이들의 조합을 포함한다.
DNA-표적화 세그먼트
DNA-표적화 RNA("crRNA")의 DNA-표적화 세그먼트(또는 "DNA-표적화 서열")은 표적 DNA 내의 특이적 서열에 상보적인 뉴클레오타이드 서열(표적 DNA의 상보적 가닥)을 포함한다.
환언하면, 당해 DNA-표적화 RNA의 DNA-표적화 세그먼트는 하이브리드화(즉, 염기 짝짓기)를 통해 서열-특이적 방식으로 표적 DNA와 상호작용한다. 이와 같이, DNA-표적화 세그먼트의 뉴클레오타이드 서열은 변할 수 있고, DNA-표적화 RNA 및 표적 DNA가 상호작용하는 표적 DNA 내의 위치를 결정할 수 있다. 당해 DNA-표적화 RNA의 DNA-표적화 세그먼트는 변형되어(예를 들면, 유전공학에 의해) 표적 DNA 내의 임의의 목적하는 서열로 하이브리드화할 수 있다.
DNA-표적화 세그먼트는 약 12 뉴클레오타이드 내지 약 100 개의 뉴클레오타이드의 길이를 가질 수 있다. 예를 들면, DNA-표적화 세그먼트는 약 12 뉴클레오타이드(nt) 내지 약 80 nt, 약 12 nt 내지 약 50 nt, 약 12 nt 내지 약 40 nt, 약 12 nt 내지 약 30 nt, 약 12 nt 내지 약 25 nt, 약 12 nt 내지 약 20 nt 또는 약 12 nt 내지 약 19 nt의 길이를 가질 수 있다. 예를 들면, DNA-표적화 세그먼트는 약 19 nt 내지 약 20 nt, 약 19 nt 내지 약 25 nt, 약 19 nt 내지 약 30 nt, 약 19 nt 내지 약 35 nt, 약 19 nt 내지 약 40 nt, 약 19 nt 내지 약 45 nt, 약 19 nt 내지 약 50 nt, 약 19 nt 내지 약 60 nt, 약 19 nt 내지 약 70 nt, 약 19 nt 내지 약 80 nt, 약 19 nt 내지 약 90 nt, 약 19 nt 내지 약 100 nt, 약 20 nt 내지 약 25 nt, 약 20 nt 내지 약 30 nt, 약 20 nt 내지 약 35 nt, 약 20 nt 내지 약 40 nt, 약 20 nt 내지 약 45 nt, 약 20 nt 내지 약 50 nt, 약 20 nt 내지 약 60 nt, 약 20 nt 내지 약 70 nt, 약 20 nt 내지 약 80 nt, 약 20 nt 내지 약 90 nt 또는 약 20 nt 내지 약 100 nt의 길이를 가질 수 있다.
표적 DNA의 뉴클레오타이드 서열(표적 서열)에 상보적인 DNA-표적화 세그먼트의 뉴클레오타이드 서열(DNA-표적화 서열)은 적어도 약 12 nt의 길이를 가질 수 있다. 예를 들면, 표적 DNA의 표적 서열에 상보적인 DNA-표적화 세그먼트의 DNA-표적화 서열은 적어도 약 12 nt, 적어도 약 15 nt, 적어도 약 18 nt, 적어도 약 19 nt, 적어도 약 20 nt, 적어도 약 25 nt, 적어도 약 30 nt, 적어도 약 35 nt 또는 적어도 약 40 nt의 길이를 가질 수 있다. 예를 들면, 표적 DNA의 표적 서열에 상보적인 DNA-표적화 세그먼트의 DNA-표적화 서열은 약 12 뉴클레오타이드 (nt) 내지 약 80 nt, 약 12 nt 내지 약 50nt, 약 12 nt 내지 약 45 nt, 약 12 nt 내지 약 40 nt, 약 12 nt 내지 약 35 nt, 약 12 nt 내지 약 30 nt, 약 12 nt 내지 약 25 nt, 약 12 nt 내지 약 20 nt, 약 12 nt 내지 약 19 nt, 약 19 nt 내지 약 20 nt, 약 19 nt 내지 약 25 nt, 약 19 nt 내지 약 30 nt, 약 19 nt 내지 약 35 nt, 약 19 nt 내지 약 40 nt, 약 19 nt 내지 약 45 nt, 약 19 nt 내지 약 50 nt, 약 19 nt 내지 약 60 nt, 약 20 nt 내지 약 25 nt, 약 20 nt 내지 약 30 nt, 약 20 nt 내지 약 35 nt, 약 20 nt 내지 약 40 nt, 약 20 nt 내지 약 45 nt, 약 20 nt 내지 약 50 nt, 또는 약 20 nt 내지 약 60 nt의 길이를 가질 수 있다. 표적 DNA의 뉴클레오타이드 서열(표적 서열)에 상보적인 DNA-표적화 세그먼트의 뉴클레오타이드 서열(DNA-표적화 서열)은 적어도 약 12 nt의 길이를 가질 수 있다.
일부 경우에서, 표적 DNA의 표적 서열에 상보적인 DNA-표적화 세그먼트의 DNA-표적화 서열은 길이 20 개의 뉴클레오타이드이다. 일부 경우에서, 표적 DNA의 표적 서열에 상보적인 DNA-표적화 세그먼트의 DNA-표적화 서열은 길이 19 뉴클레오타이드이다.
DNA-표적화 세그먼트의 DNA-표적화 서열 및 표적 DNA의 표적 서열 사이의 퍼센트 상보성은 적어도 60%(예: 적어도 65%, 적어도 70%, 적어도 75%, 적어도 80%, 적어도 85%, 적어도 90%, 적어도 95%, 적어도 97%, 적어도 98%, 적어도 99% 또는 100%)일 수 있다. 일부 경우에서, DNA-표적화 세그먼트의 DNA-표적화 서열 및 표적 DNA의 표적 서열 사이의 퍼센트 상보성은 표적 DNA의 상보성 가닥의 표적 서열의 7 인접 5'-대부분 뉴클레오타이드에 대해 100%이다. 일부 경우에서, DNA-표적화 세그먼트의 DNA-표적화 서열 및 표적 DNA의 표적 서열 사이의 퍼센트 상보성은 약 20 인접 뉴클레오타이드에 대해 적어도 60%이다. 일부 경우에서, DNA-표적화 세그먼트의 DNA-표적화 서열 및 표적 DNA의 표적 서열 사이의 퍼센트 상보성은 표적 DNA의 상보적 가닥의 표적 서열의 14 인접 5'-대부분 뉴클레오타이드에 대해 100%이고, 나머지에 대해 0%만큼 낮다. 이와 같은 경우에, DNA-표적화 서열은 길이 14 뉴클레오타이드인 것으로 간주될 수 있다. 일부 경우에서, DNA-표적화 세그먼트의 DNA-표적화 서열 및 표적 DNA의 표적 서열 사이의 퍼센트 상보성은 표적 DNA의 상보적 가닥의 표적 서열의 7 인접 5'-대부분 뉴클레오타이드에 대해 100%이고, 나머지에 대해 0%만큼 낮다. 이와 같은 경우에, DNA-표적화 서열은 길이 7 뉴클레오타이드인 것으로 간주될 수 있다.
단백질-결합 세그먼트
DNA-표적화 RNA의 단백질-결합 세그먼트(즉, "단백질-결합 서열")은 변이체 부위 지향적 폴리펩타이드와 상호작용한다. 변이체 Cas9 부위 지향적 폴리펩타이드가 DNA-표적화 RNA와 함께 표적 DNA에 결합하면, 표적 DNA의 전사가 감소된다.
DNA-표적화 RNA의 단백질-결합 세그먼트는 서로 하이브리드화하여 이중 가닥 RNA 듀플렉스(dsRNA 듀플렉스)를 형성하는 뉴클레오타이드의 두 개의 상보적 스트레치를 포함한다.
본 명세서의 DNA-표적화 RNA의 단백질-결합 세그먼트는 서로 상보적이고, 개입 뉴클레오타이드로 공유 결합되고(예: 단일-분자 DNA-표적화 RNA의 경우에)("링커" 또는 "링커 뉴클레오타이드"), 하이브리드화하여 단백질-결합 세그먼트의 이중 가닥 RNA 듀플렉스(dsRNA 듀플렉스, 또는 "dCas9-결합 헤어핀")를 형성하여 줄기-루프 구조를 초래하는 뉴클레오타이드의 두 스트레치(타겟터-RNA 및 활성제-RNA)를 포함한다. 줄기-루프 구조는 도 39A에 도식적으로 도시된다. 타겟터-RNA 및 활성제-RNA는 타겟터-RNA의 3' 말단 및 활성제-RNA의 5' 말단을 통해 공유 결합될 수 있다. 대안적으로, 타겟터-RNA 및 활성제-RNA는 타겟터-RNA의 5' 말단 및 활성제-RNA의 3' 말단을 통해 공유 결합될 수 있다.
단백질-결합 세그먼트는 약 10 개의 뉴클레오타이드 내지 약 100 개의 뉴클레오타이드, 예를 들면, 약 10 개의 뉴클레오타이드 (nt) 내지 약 20 nt, 약 20 nt 내지 약 30 nt, 약 30 nt 내지 약 40 nt, 약 40 nt 내지 약 50 nt, 약 50 nt 내지 약 60 nt, 약 60 nt 내지 약 70 nt, 약 70 nt 내지 약 80 nt, 약 80 nt 내지 약 90 nt, 또는 약 90 nt 내지 약 100 nt의 길이를 가질 수 있다. 예를 들면, 단백질-결합 세그먼트는 약 15 개의 뉴클레오타이드 (nt) 내지 약 80 nt, 약 15 nt 내지 약 50 nt, 약 15 nt 내지 약 40 nt, 약 15 nt 내지 약 30 nt 또는 약 15 nt 내지 약 25 nt의 길이를 가질 수 있다.
단백질-결합 세그먼트의 dsRNA 듀플렉스는 약 6 염기쌍(bp) 내지 약 50bp의 길이를 가질 수 있다. 예를 들면, 단백질-결합 세그먼트의 dsRNA 듀플렉스는 약 6 bp 내지 약 40 bp, 약 6 bp 내지 약 30bp, 약 6 bp 내지 약 25 bp, 약 6 bp 내지 약 20 bp, 약 6 bp 내지 약 15 bp, 약 8 bp 내지 약 40 bp, 약 8 bp 내지 약 30bp, 약 8 bp 내지 약 25 bp, 약 8 bp 내지 약 20 bp 또는 약 8 bp 내지 약 15 bp의 길이를 가질 수 있다. 예를 들면, 단백질-결합 세그먼트의 dsRNA 듀플렉스는 약 8 bp 내지 약 10 bp, 약 10 bp 내지 약 15 bp, 약 15 bp 내지 약 18 bp, 약 18 bp 내지 약 20 bp, 약 20 bp 내지 약 25 bp, 약 25 bp 내지 약 30 bp, 약 30 bp 내지 약 35 bp, 약 35 bp 내지 약 40 bp 또는 약 40 bp 내지 약 50 bp의 길이를 가질 수 있다. 일부 구현예에서, 단백질-결합 세그먼트의 dsRNA 듀플렉스는 36 염기쌍의 길이를 가질 수 있다. 하이브리드화하여 단백질-결합 세그먼트의 dsRNA 듀플렉스를 형성하는 뉴클레오타이드 서열 사이의 퍼센트 상보성은 적어도 약 60%일 수 있다. 예를 들면, 하이브리드화하여 단백질-결합 세그먼트의 dsRNA 듀플렉스를 형성하는 뉴클레오타이드 서열 사이의 퍼센트 상보성은 적어도 약 65%, 적어도 약 70%, 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 98% 또는 적어도 약 99%일 수 있다. 일부 경우에서, 하이브리드화하여 단백질-결합 세그먼트의 dsRNA 듀플렉스를 형성하는 뉴클레오타이드 서열 사이의 퍼센트 상보성은 100%이다.
링커는 약 3 뉴클레오타이드 내지 약 100 개의 뉴클레오타이드의 길이를 가질 수 있다. 예를 들면, 링커는 약 3 뉴클레오타이드 (nt) 내지 약 90 nt, 약 3 뉴클레오타이드 (nt) 내지 약 80 nt, 약 3 뉴클레오타이드 (nt) 내지 약 70 nt, 약 3 뉴클레오타이드 (nt) 내지 약 60 nt, 약 3 뉴클레오타이드 (nt) 내지 약 50 nt, 약 3 뉴클레오타이드 (nt) 내지 약 40 nt, 약 3 뉴클레오타이드 (nt) 내지 약 30 nt, 약 3 뉴클레오타이드 (nt) 내지 약 20 nt 또는 약 3 뉴클레오타이드 (nt) 내지 약 10 nt의 길이를 가질 수 있다. 예를 들면, 링커는 약 3 nt 내지 약 5 nt, 약 5 nt 내지 약 10 nt, 약 10 nt 내지 약 15 nt, 약 15 nt 내지 약 20 nt, 약 20 nt 내지 약 25 nt, 약 25 nt 내지 약 30 nt, 약 30 nt 내지 약 35 nt, 약 35 nt 내지 약 40 nt, 약 40 nt 내지 약 50 nt, 약 50 nt 내지 약 60 nt, 약 60 nt 내지 약 70 nt, 약 70 nt 내지 약 80 nt, 약 80 nt 내지 약 90 nt 또는 약 90 nt 내지 약 100 nt의 길이를 가질 수 있다. 일부 구현예에서, DNA-표적화 RNA의 링커는 4 nt이다.
적당한 단백질-결합 세그먼트(즉, dCas9 핸들)에 포함될 수 있는 뉴클레오타이드 서열의 비제한적인 예는 서열번호 563-682(예를 들어, 도 8 및 도 9 참조)에 나타낸다.
일부 경우에서, 적당한 단백질-결합 세그먼트는 상기 열거된 서열 중의 임의의 하나로부터 1, 2, 3, 4 또는 5개 뉴클레오타이드가 상이한 뉴클레오타이드 서열을 포함한다.
안정성 제어 서열(예: 전사 종결자 세그먼트)
안정성 제어 서열은 RNA(예: DNA-표적화 RNA, 타겟터-RNA, 활성제-RNA 등)의 안정성에 영향을 미친다. 적당한 안정성 제어 서열의 일례는 전사 종결자 세그먼트(즉, 전사 종결 서열)이다. 당해 DNA-표적화 RNA의 전사 종결자 세그먼트는 약 10 개의 뉴클레오타이드 내지 약 100 개의 뉴클레오타이드, 예를 들면, 약 10 개의 뉴클레오타이드 (nt) 내지 약 20 nt, 약 20 nt 내지 약 30 nt, 약 30 nt 내지 약 40 nt, 약 40 nt 내지 약 50 nt, 약 50 nt 내지 약 60 nt, 약 60 nt 내지 약 70 nt, 약 70 nt 내지 약 80 nt, 약 80 nt 내지 약 90 nt 또는 약 90 nt 내지 약 100 nt의 총 길이를 가질 수 있다. 예를 들면, 전사 종결자 세그먼트는 약 15 개의 뉴클레오타이드 (nt) 내지 약 80 nt, 약 15 nt 내지 약 50 nt, 약 15 nt 내지 약 40 nt, 약 15 nt 내지 약 30 nt 또는 약 15 nt 내지 약 25 nt의 길이를 가질 수 있다.
일부 경우에서, 전사 종결 서열은 진핵 세포에서 기능성인 것이다. 일부 경우에서, 전사 종결 서열은 원핵 세포에서 기능성인 것이다.
안정성 제어 서열(예: 전사 종결 세그먼트, 또는 증가된 안정성을 제공하기 위한 DNA-표적화 RNA의 임의의 세그먼트)에 포함될 수 있는 뉴클레오타이드 서열의 비제한적인 예는 서열번호 683-696에 나타낸 서열, 예를 들면, 5'-UAAUCCCACAGCCGCCAGUUCCGCUGGCGGCAUUUU-5'(서열번호 795)(Rho-독립적 trp 종결 부위)를 포함한다.
추가의 서열
일부 구현예에서, DNA-표적화 RNA는 5' 또는 3' 말단에서 적어도 하나의 추가의 세그먼트를 포함한다. 예를 들면, 적합한 추가의 세그먼트는 5' 캡(예: 7-메틸구아닐레이트 캡(m7G)); 3' 폴리아데닐화 꼬리(즉, 3' 폴리(A) 꼬리); 리보스위치 서열(예: 단백질 및/또는 단백질 복합체에 의한 조절된 안정성 및/또는 조절된 접근성을 허용하는); dsRNA 듀플렉스(즉, 헤어핀)를 형성하는 서열; RNA를 준세포 위치(예: 핵, 미토콘드리아, 엽록체 등)에서 표적화하는 서열; 추적(예: 형광성 분자에 직접 접합, 형광 검출을 촉진시키는 잔기에 접합, 형광 검출을 허용하는 서열 등)을 제공하는 변형 또는 서열; 단백질(예: 전사 활성제, 전사 억제인자, DNA 메틸트랜스퍼라제, DNA 데메틸라제, 히스톤 아세틸트랜스퍼라제, 히스톤 데아세틸라제 등을 포함하는 DNA에 작용하는 단백질)에 결합 부위를 제공하는 변형 또는 서열; 증가된, 감소된 및/또는 제어가능한 안정성을 제공하는 변형 또는 서열; 및 이들의 조합을 포함할 수 있다.
다중 동시 DNA-표적화 RNA
일부 구현예에서, 다중 DNA-표적화 RNA는 동일 표적 DNA 상 또는 상이한 표적 DNA 상의 상이한 위치에서 전사를 동시에 조절하기 위해 동일 세포에 동시에 사용된다. 일부 구현예에서, 2개 이상의 DNA-표적화 RNA는 동일한 유전자 또는 전사체 또는 유전자 좌를 표적화한다. 일부 구현예에서, 2개 이상의 DNA-표적화 RNA는 상이한 관련 없는 유전자 좌를 표적화한다. 일부 구현예에서, 2개 이상의 DNA-표적화 RNA는 상이하지만 관련있는 유전자 좌를 표적화한다.
DNA-표적화 RNA가 작고 강력하기 때문에, 그들은 동일 발현 벡터 상에 동시에 존재할 수 있고, 심지어 그렇게 목적하는 경우, 동일한 전사 제어하에 존재할 수 있다. 일부 구현예에서, 2개 이상(예: 3개 이상, 4개 이상, 5개 이상, 10개 이상, 15개 이상, 20개 이상, 25개 이상, 30개 이상, 35개 이상, 40개 이상, 45개 이상 또는 50개 이상)의 DNA-표적화 RNA가 (동일하거나 상이한 벡터로부터) 표적 세포에서 동시에 발현된다. 발현된 DNA-표적화 RNA는 상이한 박테리아, 예를 들면, 에스. 파이오제네스, 에스. 서모필러스, 엘. 이노쿠아 및 엔. 메닌자이티디스로부터 dCas9 단백질에 의해 상이하게 인식될 수 있다.
다중 DNA-표적화 RNA를 발현시키기 위해, Csy4 엔도리보뉴클레아제로 매개된 인공 RNA 처리 시스템이 사용될 수 있다. 다중 DNA-표적화 RNA는 전구체 전사체(예: U6 프로모터로부터 발현됨) 상의 탠덤 어레이에 연결될 수 있고, Csy4-특이적 RNA 서열에 의해 분리될 수 있다. 공-발현된 Csy4 단백질은 전구체 전사체를 다중 DNA-표적화 RNA로 절단한다. RNA 처리 시스템을 사용하는 이점은 다음을 포함한다: 첫째, 다중 프로모터를 사용할 필요가 없고; 둘째, 모든 DNA-표적화 RNA가 전구체 전사체로부터 처리되기 때문에, 그들의 농도는 유사한 dCas9-결합을 위해 표준화된다.
Csy4는 박테리아 슈도모나스 에어루기노사로부터 유도된 작은 엔도리보뉴클레아제(RNase) 단백질이다. Csy4는 특이적으로 최소 17-bp RNA 헤어핀을 인식하고, 신속하고(< 1분) 매우 효율적인(> 99.9%) RNA 절단을 나타낸다. 대부분의 RNase와 달리, 절단된 RNA 단편은 안정하고 기능적으로 활성으로 잔류한다. Csy4-기반 RNA 절단은 인공 RNA 처리 시스템 속에서 용도변경될 수 있다. 이 시스템에서, 17-bp RNA 헤어핀을 단일 프로모터로부터 전구체 전사체로서 전사되는 다중 RNA 단편 사이에 삽입한다. Csy4의 공-발현은 개별적 RNA 단편을 생성하는데 효과적이다.
부위 지향적 폴리펩타이드
상기에서 언급된 바와 같이, 당해 DNA-표적화 RNA 및 변이체 Cas9 부위 지향적 폴리펩타이드는 복합체를 형성한다. DNA-표적화 RNA는 표적 DNA의 서열에 상보적인 뉴클레오타이드 서열을 포함시킴으로써 복합체에 표적 특이성을 제공한다.
변이체 Cas9 부위 지향적 폴리펩타이드는 감소된 엔도데옥시리보뉴클레아제 활성을 갖는다. 예를 들면, 본 명세서의 전사 조절 방법에 사용하기에 적당한 변이체 Cas9 부위 지향적 폴리펩타이드는 야생형 Cas9 폴리펩타이드, 예를 들면, 도 3에 도시된 바와 같은 아미노산 서열(서열번호 8)을 포함하는 야생형 Cas9 폴리펩타이드의 엔도데옥시리보뉴클레아제 활성의 약 20% 미만, 약 15% 미만, 약 10% 미만, 약 5% 미만, 약 1% 미만 또는 약 0.1% 미만을 나타낸다. 일부 구현예에서, 변이체 Cas9 부위 지향적 폴리펩타이드는 실질적으로 검출가능한 엔도데옥시리보뉴클레아제 활성을 갖지 않는다. 일부 구현예에서, 부위 지향적 폴리펩타이드가 감소된 촉매 활성을 갖는 경우(예: Cas9 단백질이 D10, G12, G17, E762, H840, N854, N863, H982, H983, A984, D986, 및/또는 A987 돌연변이, 예를 들면, D10A, G12A, G17A, E762A, H840A, N854A, N863A, H982A, H983A, A984A, 및/또는 D986A를 가질 경우), 폴리펩타이드는 DNA-표적화 RNA와 상호작용하는 능력을 유지하는 한, (그것은 DNA-표적화 RNA에 의해 표적 DNA 서열로 여전히 유도되기 때문에) 부위 특이적 방식으로 표적 DNA에 결합할 수 있다.
일부 경우에서, 적당한 변이체 Cas9 부위 지향적 폴리펩타이드는 도 3에 묘사된 Cas9/Csn1 아미노산 서열(서열번호 8)의 아미노산 7-166 또는 731-1003 또는 아미노산 서열 서열번호 1-256 및 795-1346 중의 임의의 하나의 상응하는 부분과 적어도 약 75%, 적어도 약 80%, 적어도 약 85%, 적어도 약 90%, 적어도 약 95%, 적어도 약 99% 또는 100% 아미노산 서열 동일성을 갖는 아미노산 서열을 포함한다.
일부 경우에서, 변이체 Cas9 부위 지향적 폴리펩타이드는 표적 DNA의 상보적 가닥을 절단할 수 있지만, 표적 DNA의 비-상보적 가닥을 절단하는 능력은 감소된다. 예를 들면, 변이체 Cas9 부위 지향적 폴리펩타이드는 RuvC 도메인(예: 도 3의 "도메인 1")의 기능을 감소시키는 돌연변이(아미노산 치환)를 가질 수 있다. 비제한적인 예로서, 일부 경우에서, 변이체 Cas9 부위 지향적 폴리펩타이드는 도 3에 묘사된 아미노산 서열의 D10A(아스파테이트 대 알라닌) 돌연변이(또는 서열번호 1-256 및 795-1346에 나타낸 임의의 아미노산 서열의 상응하는 돌연변이)이다.
일부 경우에서, 변이체 Cas9 부위 지향적 폴리펩타이드는 표적 DNA의 비-상보적 가닥을 절단할 수 있지만, 표적 DNA의 상보적 가닥을 절단하는 능력은 감소된다. 예를 들면, 변이체 Cas9 부위 지향적 폴리펩타이드는 HNH 도메인(RuvC/HNH/RuvC 도메인 모티프, 도 3의 "도메인 2")의 기능을 감소시키는 돌연변이(아미노산 치환)를 가질 수 있다. 비제한적인 예로서, 일부 경우에서, 변이체 Cas9 부위 지향적 폴리펩타이드는 H840A(서열번호 8의 아미노산 위치 840에서 히스티딘 대 알라닌) 또는 서열번호 1-256 및 795-1346에 나타낸 임의의 아미노산 서열의 상응하는 돌연변이)이다.
일부 경우에서, 변이체 Cas9 부위 지향적 폴리펩타이드는 표적 DNA의 상보적 및 비-상보적 가닥 둘 다를 절단하는 능력이 감소된다. 비제한적인 예로서, 일부 경우에서, 변이체 Cas9 부위 지향적 폴리펩타이드는 도 3에 묘사된 아미노산 서열의 D10A 및 H840A 돌연변이 모두(또는 서열번호 1-256 및 795-1346에 나타낸 임의의 아미노산 서열의 상응하는 돌연변이)를 포함한다.
기타 잔기는 돌연변이되어 동일한 효과를 달성할 수 있다(즉, 하나 또는 다른 뉴클레아제 부분을 불활성화할 수 있다). 비제한적인 예로서, 잔기 D10, G12, G17, E762, H840, N854, N863, H982, H983, A984, D986 및/또는 A987(또는 서열번호 1-256 및 795-1346로서 나타낸 임의의 단백질의 상응하는 돌연변이)은 변경될 수 있다(즉, 치환될 수 있다)(참조: Cas9 아미노산 잔기의 보존에 관한 추가의 정보를 위해 도 3, 도 5, 도 11a 및 표 1). 또한, 알라닌 치환 이외의 돌연변이가 적당하다.
일부 경우에, 변이체 Cas9 부위 지향적 폴리펩타이드는 융합 폴리펩타이드("변이체 Cas9 융합 폴리펩타이드"), 즉 i) 변이체 Cas9 부위 지향적 폴리펩타이드; 및 b) 공유 결합된 이종기원 폴리펩타이드(또한 "융합 파트너"로서 칭명됨)를 포함하는 융합 폴리펩타이드이다.
이종기원 폴리펩타이드는 또한 변이체 Cas9 융합 폴리펩타이드에 의해 나타나는 활성(예: 효소 활성)(예: 메틸트랜스퍼라제 활성, 아세틸트랜스퍼라제 활성, 키나제 활성, 우비퀴틴화 활성 등)을 나타낼 수 있다. 이종기원 핵산 서열은 또 하나의 핵산 서열에 (예를 들면, 유전공학에 의해) 결합하여 키메라 폴리펩타이드를 인코딩하는 키메라 뉴클레오타이드 서열을 생성할 수 있다. 일부 구현예에서, 변이체 Cas9 융합 폴리펩타이드는 준세포 국재화를 제공하는 이종기원 서열로 변이체 Cas9 폴리펩타이드를 융합시킴으로써 생성된다(즉, 이종기원 서열은 준세포 국재화 서열, 예를 들면, 핵에 표적화하기 위한 핵 국재화 신호(NLS); 미토콘드리아에 표적화하기 위한 미토콘드리아 국재화 신호; 엽록체에 표적화하기 위한 엽록체 국재화 신호; ER 체류 신호 등이다). 일부 구현예에서, 이종기원 서열은 추적 및 정제의 용이성을 위해 택(즉, 이종기원 서열은 검출가능한 라벨이다)(예: 형광 단백질, 예를 들면, 그린 형광 단백질(GFP), YFP, RFP, CFP, mCherry, tdTomato 등; 히스티딘 택, 예를 들면, 6XHis 택; 헤마글루티닌(HA) 택; FLAG tag; Myc 택 등)을 제공할 수 있다. 일부 구현예에서, 이종기원 서열은 증가되거나 감소된 안정성을 제공할 수 있다(즉, 이종기원 서열은 일부 경우에서 제어가능한 안정성 제어 펩타이드, 예를 들면, 데그론(예: 온도 민감성 또는 약물 제어가능한 데그론 서열, 이하 참조)이다). 일부 구현예에서, 이종기원 서열은 표적 DNA로부터 증가되거나 감소된 전사를 제공할 수 있다(즉, 이종기원 서열은 전사 조절 서열, 예를 들면, 전사 인자/활성제 또는 이의 단편, 전사 인자/활성제를 모집하는 단백질 또는 이의 단편, 전사 억제인자 또는 이의 단편, 전사 억제인자, 소분자/약물 반응성 전사 조절제를 모집하는 단백질 또는 이의 단편 등이다). 일부 구현예에서, 이종기원 서열은 결합 도메인을 제공할 수 있다(즉, 이종기원 서열은, 예를 들면, 목적하는 또 하나의 단백질, 예를 들면, DNA 또는 히스톤 변형 단백질, 전사 인자 또는 전사 억제인자, 모집 단백질 등에 결합하는 키메라 dCas9 폴리펩타이드의 능력을 제공하기 위한 단백질 결합 서열이다).
증가되거나 감소된 안정성을 제공하는 적당한 융합 파트너는 데그론 서열을 포함하지만, 이에 국한되지 않는다. 데그론은 그들이 일부인 단백질의 안정성을 제어하는 아미노산 서열인 것으로 당해 기술 분야의 숙련가에 의해 용이하게 이해된다. 예를 들면, 데그론 서열을 포함하는 단백질의 안정성은 데그론 서열에 의해 적어도 부분적으로 제어된다. 일부 경우에서, 적당한 데그론은, 데그론이 실험적 제어와 관계없이 단백질 안정성에 대한 이의 영향을 발휘하도록 구성적이다(즉, 데그론은 약물 유도성, 온도 유도성 등이 아니다). 일부 경우에서, 데그론은 변이체 Cas9 폴리펩타이드가 목적하는 조건에 따라 "온"(즉, 안정) "오프"(즉, 불안정, 퇴화)될 수 있도록 제어가능한 안정성을 갖는 변이체 Cas9 폴리펩타이드를 제공한다. 예를 들면, 데그론이 온도 민감성 데그론일 경우, 변이체 Cas9 폴리펩타이드는 임계 온도 이하(예: 42℃, 41℃, 40℃, 39℃, 38℃, 37℃, 36℃, 35℃, 34℃, 33℃, 32℃, 31℃, 30℃ 등)에서 기능성(즉, "온", 안정)일 수 있지만, 임계 온도 이상에서는 비기능성(즉, "오프", 퇴화됨)일 수 있다. 또 하나의 예로서, 데그론이 약물 유도성 데그론일 경우, 약물의 존재 또는 부재는 단백질을 "오프"(즉, 불안정) 상태에서 "온"(즉, 안정) 상태로 또는 그 반대로 작동시킬 수 있다. 예시적인 약물 유도성 데그론은 FKBP12 단백질로부터 유도된다. 데그론의 안정성은 데그론에 결합하는 소분자의 존재 또는 부재로 제어된다.
적당한 데그론의 예는 쉴드-1, DHFR, 옥신 및/또는 온도에 의해 조절된 데그론을 포함하지만, 이에 국한되지 않는다. 적당한 데그론의 비제한적인 에는 당해 기술 분야에 공지되어 있다(참조: Dohmen 등, Science, 1994. 263(5151): p. 1273-1276: Heat-inducible degron: a method for constructing temperature-sensitive mutants; Schoeber 등, Am J Physiol Renal Physiol. 2009 Jan;296(1):F204-11 : Conditional fast expression and function of multimeric TRPV5 channels using Shield-1 ; Chu 등, Bioorg Med Chem Lett. 2008 Nov 15;18(22):5941-4: Recent progress with FKBP-derived destabilizing domains ; Kanemaki, Pflugers Arch. 2012 Dec 28: Frontiers of protein expression control with conditional degrons; Yang 등, Mol Cell. 2012 Nov 30;48(4):487-8: Titivated for destruction: the methyl degron; Barbour 등, Biosci Rep. 2013 Jan 18;33(1).: Characterization of the bipartite degron that regulates ubiquitin-independent degradation of thymidylate synthase; 및 Greussing 등, J Vis Exp. 2012 Nov 10;(69): Monitoring of ubiquitin-proteasome activity in living cells using a Degron (dgn)-destabilized green fluorescent protein (GFP)-based reporter protein; 이들 모두는 본원에 참조로 인용된다).
예시적인 데그론 서열은 충분히 특성화되어 있고, 세포 및 동물 모두에서 시험되고 있다. 따라서, 데그론 서열에 dCas9의 융합은 "조정성" 및 "유도성" dCas9 폴리펩타이드를 생성한다. 본원에서 기재된 임의의 융합 파트너는 임의의 바람직한 조합으로 사용될 수 있다. 이 점을 설명하기 위한 하나의 비제한적인 예로서, dCas9 융합 단백질은 검출용 YFP 서열, 안정성을 위한 데그론 서열 및 표적 DNA의 전사를 증가시키기 위한 전사 활성제 서열을 포함할 수 있다. 또한, dCas9 융합 단백질에 사용될 수 있는 융합 파트너의 수는 비제한된다. 일부 경우에서, dCas9 융합 단백질은 하나 이상(예: 둘 이상, 셋 이상, 넷 이상 또는 다섯 이상)의 이종기원 서열을 포함한다.
적당한 융합 파트너는 메틸트랜스퍼라제 활성, 데메틸라제 활성, 아세틸트랜스퍼라제 활성, 데아세틸라제 활성, 키나제 활성, 포스파타제 활성, 우비퀴틴 리가제 활성, 탈우비퀴틴화 활성, 아데닐화 활성, 탈아데닐화 활성, SUMO닐화 활성, 탈SUMO닐화 활성, 리보실화 활성, 탈리보실화 활성, 미리스토일화 활성 또는 탈미리스토일화 활성을 제공하는 폴리펩타이드를 포함하지만, 이에 국한되지 않고, 이중 임의의 것은 DNA를 직접 변형시키는데(예: DNA의 메틸화) 또는 DNA 관련 폴리펩타이드(예: 히스톤 또는 DNA 결합 단백질)를 변형시키는데 지시될 수 있다. 추가의 적당한 융합 파트너는 주변 모집(예: 라민 A, 라민 B 등)을 제공하는 경계 요소(예: CTCF), 단백질 및 이의 단편, 및 단백질 도킹 요소(예: FKBP/FRB, Pil1/Aby1 등)를 포함하지만, 이에 제한되지 않는다.
당해 변이체 Cas9 부위 지향적 폴리펩타이드를 위한 다양한 추가의 적당한 융합 파트너(또는 이의 단편)의 예는 도 54에 열거된 것들을 포함하지만, 이에 국한되지 않는다.
일부 구현예에서, 당해 부위 지향적 변형 폴리펩타이드는 코돈-최적화될 수 있다. 이러한 유형의 최적화는 당해 기술 분야에 공지되어 있고, 동일 단백질을 인코딩하면서 의도된 숙주 유기체 또는 세포의 코돈 선호를 모방하는 외래 유도된 DNA의 돌연변이를 수반한다. 따라서, 코돈은 변하지만, 인코딩된 단백질은 변하지 않는다. 예를 들면, 의도된 표적 세포가 인간 세포일 경우, 인간 코돈 최적화된 dCas9 (또는 dCas9 변이체)는 적당한 부위 지향적 변형 폴리펩타이드일 것이다. 또 하나의 비제한적인 예로서, 의도된 숙주 세포가 마우스 세포이면, 마우스 코돈 최적화된 Cas9 (또는 변이체, 예를 들면, 효소적 불활성 변이체)는 적당한 Cas9 부위 지향적 폴리펩타이드일 것이다. 코돈 최적화가 필요하지 않는 반면, 이는 특정 경우에 허용되고, 바람직할 수 있다.
숙주 세포
전사를 조절하기 위한 본 명세서의 방법을 사용하여 생체내 및/또는 생체외 및/또는 시험관내 유사분열 또는 후-유사분열 세포에서 전사 조절을 유도할 수 있다. DNA-표적화 RNA가 표적 DNA로 하이브리드화함으로써 특이성을 제공하기 때문에, 유사분열 및/또는 후-유사분열 세포는 임의의 다양한 숙주 세포일 수 있고, 여기서 적당한 숙주 세포는 박테리아 세포; 고세균 세포; 단일-세포 진핵 유기체; 식물 세포; 조류 세포, 예를 들면, 보트라이오코쿠스 브라우니이(Botryococcus braunii), 클라마이도모나스 레인하르드티이(Chlamydomonas reinhardtii), 난노클로롭시스 가디타나(Nannochloropsis gaditana), 클로렐라 파이레노이도사( Chlorella pyrenoidosa ), 사르가섬 파텐스 ( Sargassum patens ), C. 아가르드(C. agardh) 등; 진균 세포; 동물 세포; 무척추동물 동물(예: 곤충, 자포동물, 극피동물, 선충 등)로부터의 세포; 진핵 기생충(예: 말라리아 기생충, 예를 들면, 플라스모디엄 팔시파럼(Plasmodium falciparum); 연충 등); 척추동물(예: 어류, 양서류, 파충류, 새, 포유동물)로부터의 세포; 포유동물 세포, 예를 들면, 설치류 세포, 인간 세포, 비-인간 영장류 세포 등을 포함하지만, 이에 국한되지 않는다. 적당한 숙주 세포는 자연-발생 세포; 유전적으로 변형된 세포(예: 실험실에서, 예를 들면, "사람의 손"에 의해 유전적으로 변형된 세포); 및 어떤 방식으로 시험관내에서 조작된 세포를 포함하지만, 이에 국한되지 않는다. 일부 경우에서, 숙주 세포가 단리된다.
임의 유형의 세포가 목적시될 수 있다(예: 줄기 세포, 예를 들면, 배아 줄기(ES) 세포, 유도된 만능 줄기(iPS) 세포, 생식세포; 체세포, 예를 들면, 섬유아세포, 조혈 세포, 뉴런, 근육 세포, 골 세포, 간세포, 췌장 세포; 임의 단계에서 배아, 예를 들면, 1-세포, 2-세포, 4-세포, 8-세포 등의 단계 제브라피쉬 배아의 시험관내 또는 생체내 배아 세포 등). 세포는 확립된 세포주로부터일 수 있거나, 그들은 1차 세포일 수 있고, 여기서 "1차 세포", "1차 세포주" 및 "1차 배양물"은 대상체로부터 유도되어 배양물의 제한된 수의 계대, 즉 쪼갬 동안 시험관내에서 성장시키는 세포 및 세포 배양물을 의미하기 위해 본원에서 상호교환적으로 사용된다. 예를 들면, 1차 배양물은 0회, 1회, 2회, 4회, 5회, 10회 또는 15회 계대될 수 있지만, 위기 단계를 통과하기에 충분한 회수는 아닌 배양물을 포함한다. 1차 세포주는 시험관내에서 10회 미만 계대 동안 유지시킬 수 있다. 표적 세포는 많은 구현예에서, 단세포 유기체이거나, 배양물에서 성장된다.
세포가 1차 세포이면, 이러한 세포는 임의의 편리한 방법에 의해 개체로부터 채취할 수 있다. 예를 들면, 백혈구는 분리반출법, 백혈구분리반출법, 밀도 구배 분리 등에 의해 편리하게 채취할 수 있는 반면, 조직, 예를 들면, 피부, 근육, 골수, 비장, 간, 췌장, 폐, 창자, 위 등으로부터 세포는 생검에 의해 가장 편리하게 채취된다. 적당한 용액은 채취된 세포의 분산 또는 현탁을 위해 사용될 수 있다. 이러한 용액은 일반적으로 저농도, 예를 들면, 5 내지 25mM에서 허용되는 완충제와 함께 소 태아 혈청 또는 기타 자연 발생 인자로 편리하게 보충된 평형 염 용액, 예를 들면, 생리 식염수, 포스페이트 완충된 염수(PBS), 행크 평형 염 용액 등이다. 편리한 완충제는 HEPES, 포스페이트 완충제, 락테이트 완충제 등을 포함한다. 세포는 즉시 사용될 수 있거나, 그들은 긴 시간 동안 저장, 냉동될 수 있고, 해동되어 재사용될 수 있다. 이러한 경우에, 세포는 일반적으로 이러한 동결 온도에서 세포를 보존하기 위해 당해 기술 분야에서 통상 사용되는 바와 같이 10% 디메틸 설폭사이드(DMAO), 50% 혈청, 40% 완충 배지 또는 일부 다른 상기 용액에서 동결시키고, 동결된 배양 세포를 해동하기 위해 당해 기술 분야에 통상 공지된 바와 같은 방식으로 해동시킨다.
숙주 세포에 핵산의 도입
DNA-표적화 RNA, 또는 이를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산을 임의의 다양한 익히 공지된 방법에 의해 숙주 세포에 도입할 수 있다. 유사하게, 당해 방법이 숙주 세포에 변이체 Cas9 부위 지향적 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산을 도입함을 포함할 경우, 이러한 핵산은 임의의 다양한 익히 공지된 방법에 의해 숙주 세포에 도입할 수 있다.
핵산을 숙주 세포에 도입하는 방법은 당해 기술 분야에 공지되어 있고, 임의의 공지된 방법은 핵산(예: 발현 작제물)을 줄기 세포 또는 전구 세포에 도입하기 위해 사용될 수 있다. 적당한 방법은, 예를 들면, 바이러스 또는 박테리오파지 감염, 형질감염, 접합, 원형질 융합, 리포펙션, 전기천공, 인산칼슘 침전, 폴리에틸렌이민(PEI)-매개된 형질감염, DEAE-덱스트란 매개된 형질감염, 리포좀-매개된 형질감염, 입자 총 기술, 인산칼슘 침전, 직접 미세 주입, 나노입자-매개된 핵산 전달(참조: 예를 들면, Panyam 등 Adv Drug Deliv Rev. 2012 Sep 13. pii: S0169-409X(12)00283-9. doi: 10.1016/j.addr.2012.09.023) 등을 포함한다.
핵산
본 명세서는 당해 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 단리된 핵산을 제공한다. 일부 경우에서, 당해 핵산은 또한 변이체 Cas9 부위 지향적 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함한다.
일부 구현예에서, 당해 방법은 숙주 세포(또는 숙주 세포의 모집단)에 DNA-표적화 RNA 및/또는 변이체 Cas9 부위 지향적 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 하나이상의 핵산을 도입함을 포함한다. 일부 구현예에서, 표적 DNA를 포함하는 세포는 시험관내에 있다. 일부 구현예에서, 표적 DNA를 포함하는 세포는 생체내에 있다. DNA-표적화 RNA 및/또는 부위 지향적 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 적당한 핵산은 발현 벡터를 포함하고, 여기서 DNA-표적화 RNA 및/또는 부위 지향적 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 발현 벡터는 "재조합 발현 벡터"이다.
일부 구현예에서, 재조합 발현 벡터는 바이러스 작제물, 예를 들면, 재조합 아데노-연관된 바이러스 작제물(참조: 예를 들면, 미국 특허 제7,078,387호), 재조합 아데노바이러스 작제물, 재조합 렌티바이러스 작제물, 재조합 레트로바이러스 작제물 등이다.
적당한 발현 벡터는 바이러스 벡터(예: 우두 바이러스; 폴리오바이러스; 아데노바이러스(참조: 예를 들면, Li 등, Invest Opthalmol Vis Sci 35:2543 2549, 1994; Borras 등, Gene Ther 6:515 524, 1999; Li and Davidson, PNAS 92:7700 7704, 1995; Sakamoto 등, H Gene Ther 5:1088 1097, 1999; WO 94/12649, WO 93/03769; WO 93/19191; WO 94/28938; WO 95/11984 및 WO 95/00655); 아데노-연관된 바이러스(참조: 예를 들면, Ali 등, Hum Gene Ther 9:81 86, 1998, Flannery 등, PNAS 94:6916 6921, 1997; Bennett 등, Invest Opthalmol Vis Sci 38:2857 2863, 1997; Jomary 등, Gene Ther 4:683 690, 1997, Rolling 등, Hum Gene Ther 10:641 648, 1999; Ali 등, Hum Mol Genet 5:591 594, 1996; Srivastava in WO 93/09239, Samulski 등, J. Vir. (1989) 63:3822-3828; Mendelson 등, Virol. (1988) 166:154-165; 및 Flotte 등, PNAS (1993) 90:10613-10617); SV40; 단순 포진 바이러스; 인간 면역결핍 바이러스(참조: 예를 들면, Miyoshi 등, PNAS 94:10319 23, 1997; Takahashi 등, J Virol 73:7812 7816, 1999); 레트로바이러스 벡터(예: 쥣과 백혈병 바이러스, 비장 괴사 바이러스 및 레트로바이러스, 예를 들면, 라우스 육종 바이러스, 하비 육종 바이러스, 조류 백혈병 바이러스, 렌티바이러스, 인간면역 바이러스, 골수증식성 육종 바이러스 및 유선 종양 바이러스) 등을 기반으로 하는 바이러스 벡터)를 포함하지만, 이에 국한되지 않는다.
수많은 적당한 발현 벡터는 당해 기술 분야의 숙련가에게 공지되어 있고, 다수는 시판되고 있다. 다음 벡터가 예로써 제공된다: 진핵 숙주 세포의 경우: pXT1, pSG5(Stratagene), pSVK3, pBPV, pMSG 및 pSVLSV40(Pharmacia). 그러나, 임의의 기타 벡터가 숙주 세포와 양립가능한 한 사용될 수 있다.
사용된 숙주/벡터 시스템에 따라, 구성성 및 유도성 프로모터, 전사 인핸서 요소, 전사 종결자 등을 포함하는 임의의 다수의 적당한 전사 및 번역 조절 요소가 발현 벡터에 사용될 수 있다(참조: 예를 들면, Bitter 등 (1987) Methods in Enzymology, 153:516-544).
일부 구현예에서, DNA-표적화 RNA 및/또는 변이체 Cas9 부위 지향적 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열은 조절 요소, 예를 들면, 전사 조절 요소, 예를 들면, 프로모터에 작동가능하게 연결된다. 전사 조절 요소는 진핵 세포, 예를 들면, 포유동물 세포, 또는 원핵 세포(예: 세균 또는 고세균 세포)에서 기능성일 수 있다. 일부 구현예에서, DNA-표적화 RNA 및/또는 변이체 Cas9 부위 지향적 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열은 원핵 및 진핵 세포 둘 다에서 DNA-표적화 RNA 및/또는 변이체 Cas9 부위 지향적 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열의 발현을 허용하는 다중 조절 요소에 작동가능하게 연결된다.
프로모터는 구조적 활성 프로모터(즉, 구조적으로 활성/"온" 상태로 존재하는 프로모터)일 수 있고, 이는 유도성 프로모터일 수 있고(즉, 상태가 활성/"온" 또는 불활성/"오프" 상태의 프로모터는 외부 자극, 예를 들면, 특정 온도, 화합물 또는 단백질의 존재에 의해 조절된다), 이는 공간적으로 제한된 프로모터(즉, 전사 조절 요소, 인핸서 등)(즉, 조직 특이적 프로모터, 세포형 특이적 프로모터 등)일 수 있고, 이는 일시적으로 제한된 프로모터일 수 있다(즉, 프로모터는 배아 발생의 특이적 단계 동안 또는 생물학적 과정의 특이적 단계, 예를 들면, 마우스의 모낭 사이클 동안 "온" 상태 또는 "오프" 상태로 존재한다).
적당한 프로모터는 바이러스로부터 유도될 수 있고, 따라서 바이러스 프로모터로서 칭명될 수 있거나, 그들은 원핵 또는 진핵 유기체를 포함하는 임의의 유기체로부터 유도될 수 있다. 적당한 프로모터는 임의의 RNA 폴리머라제(예: pol I, pol II, pol III)에 의한 발현을 구동시키기 위해 사용될 수 있다. 예시적인 프로모터는 SV40 초기 프로모터, 마우스 유선 종양 바이러스 긴 말단 반복(LTR) 프로모터; 아데노바이러스 주요 후기 프로모터(Ad MLP); 단순 포진 바이러스(HSV) 프로모터, 사이토메갈로바이러스(CMV) 프로모터, 예를 들면, CMV 전초기 프로모터 영역(CMVIE), 라우스 육종 바이러스(RSV) 프로모터, 인간 U6 소핵 프로모터(U6)(참조: Miyagishi 등 , Nature Biotechnology 20, 497 - 500 (2002)), 증대된 U6 프로모터(예: Xia 등, Nucleic Acids Res. 2003 Sep 1;31(17)), 인간 H1 프로모터(H1) 등을 포함하지만, 이에 국한되지 않는다.
유도성 프로모터의 예는 T7 RNA 폴리머라제 프로모터, T3 RNA 폴리머라제 프로모터, 이소프로필-베타-D-티오갈락토피라노사이드(IPTG)-조절된 프로모터, 락토오스 유도된 프로모터, 열충격 프로모터, 테트라사이클린-조절된 프로모터(예: Tet-ON, Tet-OFF 등), 스테로이드-조절된 프로모터, 금속-조절된 프로모터, 에스트로겐 수용체-조절된 프로모터 등을 포함하지만, 이에 국한되지 않는다. 유도성 프로모터는 따라서 독시사이클린; RNA 폴리머라제, 예를 들면, T7 RNA 폴리머라제; 에스트로겐 수용체; 에스트로겐 수용체 융합 등을 포함하지만, 이에 국한되지 ?는 분자에 의해 조절될 수 있다.
일부 구현예에서, 프로모터는 다중-세포성 유기체에서, 프로모터가 특이적 세포의 서브 세트에서 활성(즉, "온")이도록 공간적으로 제한된 프로모터(즉, 세포형 특이적 프로모터, 조직 특이적 프로모터 등)이다. 공간적으로 제한된 프로모터는 또한 인핸서, 전사 조절 요소, 조절 서열 등으로서 칭명될 수 있다. 임의의 편리한 공간적으로 제한된 프로모터가 사용될 수 있고, 적합한 프로모터(예: 뇌 특이적 프로모터, 뉴런의 서브세트에서 발현을 구동하는 프로모터, 생식계열에서 발현을 구동하는 프로모터, 폐에서 발현을 구동하는 프로모터, 근육에서 발현을 구동하는 프로모터, 췌장의 소도세포에서 발현을 구동하는 프로모터 등)의 선택은 유기체에 좌우된다. 예를 들면, 다양한 공간적으로 제한된 프로모터는 식물, 나는 것, 벌레, 포유동물, 마우스 등에 대해 공지되어 있다. 따라서, 공간적으로 제한된 프로모터는 유기체에 따라 각종 다양한 상이한 조직 및 세포형에서 당해 부위 지향적 폴리펩타이드를 인코딩하는 핵산의 발현을 조절하기 위해 사용될 수 있다. 일부 공간적으로 제한된 프로모터는 또한 프로모터가 배아 발생의 특이적 단계 동안 또는 생물학적 과정의 특이적 단계 동안(예: 마우스의 모낭 사이클) "온" 상태 또는 "오프" 상태로 존재하도록 일시적으로 제한된다.
설명하기 위해, 공간적으로 제한된 프로모터의 예는 뉴런-특이적 프로모터, 지방세포-특이적 프로모터, 심근세포-특이적 프로모터, 평활근-특이적 프로모터, 광수용체-특이적 프로모터 등을 포함하지만, 이에 국한되지 않는다. 뉴런-특이적 공간적으로 제한된 프로모터는 뉴런-특이적 에놀라제(NSE) 프로모터(참조: 예를 들면, EMBL HSENO2, X51956); 방향족 아미노산 데카복실라제(AADC) 프로모터; 신경필라멘트 프로모터(참조: 예를 들면, GenBank HUMNFL, L04147); 시냅신 프로모터(참조: 예를 들면, GenBank HUMSYNIB, M55301); thy-1 프로모터(참조: 예를 들면, Chen 등 (1987) Cell 51:7-19; 및 Llewellyn, 등 (2010) Nat. Med. 16(10):1161-1166); 세로토닌 수용체 프로모터(참조: 예를 들면, GenBank S62283); 티로신 하이드록실라제 프로모터(TH)(참조: 예를 들면, Oh 등 (2009) Gene Ther 16:437; Sasaoka 등 (1992) Mol. Brain Res. 16:274; Boundy 등 (1998) J. Neurosci. 18:9989; 및 Kaneda 등 (1991) Neuron 6:583-594); GnRH 프로모터(참조: 예를 들면, Radovick 등 (1991) Proc. Natl. Acad. Sci. USA 88:3402-3406); L7 프로모터(참조: 예를 들면, Oberdick 등 (1990) Science 248:223-226); DNMT 프로모터(참조: 예를 들면, Bartge 등 (1988) Proc. Natl. Acad. Sci. USA 85:3648-3652); 엔케팔린 프로모터(참조: 예를 들면, Comb 등 (1988) EMBO J. 17:3793-3805); 수초 염기성 단백질(MBP) 프로모터; Ca2+-칼모둘린-의존적 단백질 키나제 II-알파(CamKIIα) 프로모터(참조: 예를 들면, Mayford 등 (1996) Proc. Natl. Acad. Sci. USA 93:13250; 및 Casanova 등 (2001) Genesis 31:37); CMV 인핸서/혈소판-유도된 성장 인자-β 프로모터(참조: 예를 들면, Liu 등 (2004) Gene Therapy 11:52-60) 등을 포함하지만, 이에 국한되지 않는다.
지방세포-특이적 공간적으로 제한된 프로모터는 aP2 유전자 프로모터/인핸서, 예를 들면, 인간 aP2 유전자의 -5.4kb 내지 +21bp의 영역(참조: 예를 들면, Tozzo 등 (1997) Endocrinol. 138:1604; Ross 등 (1990) Proc. Natl. Acad. Sci. USA 87:9590; 및 Pavjani 등 (2005) Nat. Med. 11:797); 글루코스 수송체-4(GLUT4) 프로모터(참조: 예를 들면, Knight 등 (2003) Proc. Natl. Acad. Sci. USA 100:14725); 지방산 트랜스로카제 (FAT/CD36) 프로모터(참조: 예를 들면, Kuriki 등 (2002) Biol. Pharm. Bull. 25:1476; 및 Sato 등 (2002) J. Biol. Chem. 277:15703); 스테아로일-CoA 데사투라제-1 (SCD1) 프로모터(참조: Tabor 등 (1999) J. Biol. Chem. 274:20603); 렙틴 프로모터(참조: 예를 들면, Mason 등 (1998) Endocrinol. 139:1013; 및 Chen 등 (1999) Biochem. Biophys. Res. Comm. 262:187); 아디포넥틴 프로모터(참조: 예를 들면, Kita 등 (2005) Biochem. Biophys. Res. Comm. 331:484; 및 Chakrabarti (2010) Endocrinol. 151:2408); 아딥신 프로모터(참조: 예를 들면, Platt 등 (1989) Proc. Natl. Acad. Sci. USA 86:7490); 레지스틴 프로모터(참조: 예를 들면, Seo 등 (2003) Molec. Endocrinol. 17:1522) 등을 포함하지만, 이에 국한되지 않는다.
심근세포-특이적 공간적으로 제한된 프로모터는 다음 유전자로부터 유도된 제어 서열을 포함하지만, 이에 국한되지 않는다: 미오신 경쇄-2, α-미오신 중쇄, AE3, 심장 트로포닌 C, 심장 액틴 등(참조: Franz 등 (1997) Cardiovasc. Res. 35:560-566; Robbins 등 (1995) Ann. N.Y. Acad. Sci. 752:492-505; Linn 등 (1995) Circ. Res. 76:584-591; Parmacek 등 (1994) Mol. Cell. Biol. 14:1870-1885; Hunter 등 (1993) Hypertension 22:608-617; 및 Sartorelli 등 (1992) Proc. Natl. Acad. Sci. USA 89:4047-4051).
평활근-특이적 공간적으로 제한된 프로모터는 SM22α 프로모터(참조: 예를 들면, Akyurek 등 (2000) Mol. Med. 6:983; 및 미국 특허 제7,169,874호); 스무텔린 프로모터(참조: 예를 들면, WO 2001/018048); α-평활근 액틴 프로모터 등을 포함하지만, 이에 국한되지 않는다. 예를 들면, 두 CArG 요소가 존재하는 SM22α 프로모터의 0.4 kb 영역은 혈관 평활근 세포-특이적 발현을 매개하는 것으로 나타났다(참조: 예를 들면, Kim, 등 (1997) Mol. Cell. Biol. 17, 2266-2278; Li, 등, (1996) J. Cell Biol. 132, 849-859; 및 Moessler, 등 (1996) Development 122, 2415-2425).
광수용체-특이적 공간적으로 제한된 프로모터는 로돕신 프로모터; 로돕신 키나아제 프로모터(참조: Young 등 (2003) Ophthalmol. Vis. Sci. 44:4076); 베타 포스포디에스테라제 유전자 프로모터(참조: Nicoud 등 (2007) J. Gene Med. 9:1015); 색소성 망막염 유전자 프로모터(참조: Nicoud 등 (2007) 상기와 동일); 상호광수용체 레티노이드-결합 단백질(IRBP) 유전자 인핸서(참조: Nicoud 등 (2007) 상기와 동일); IRBP 유전자 프로모터(참조: Yokoyama 등 (1992) Exp Eye Res. 55:225) 등을 포함하지만, 이에 국한되지 않는다.
라이브러리
본 명세서는 DNA-표적화 RNA의 라이브러리를 제공한다. 본 명세서는 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드를 포함하는 핵산의 라이브러리를 제공한다. DNA-표적화 RNA를 인코딩하는 뉴클레오타이드를 포함하는 핵산의 당해 라이브러리는 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드를 포함하는 재조합 발현 벡터의 라이브러리를 포함할 수 있다.
당해 라이브러리는 약 10 개별적인 멤버 내지 약 1012 개별적인 멤버를 포함할 수 있다; 예를 들면, 당해 라이브러리는 약 10 개별적인 멤버 내지 약 102 개별적인 멤버, 약 102 개별적인 멤버 내지 약 103 개별적인 멤버, 약 103 개별적인 멤버 내지 약 105 개별적인 멤버, 약 105 개별적인 멤버 내지 약 107 개별적인 멤버, 약 107 개별적인 멤버 내지 약 109 개별적인 멤버 또는 약 109 개별적인 멤버 내지 약 1012 개별적인 멤버를 포함할 수 있다.
당해 라이브러리의 "개별적인 멤버"는 DNA-표적화 RNA의 DNA 표적화 서열의 뉴클레오타이드 서열 중의 라이브러리의 다른 멤버와 상이하다. 따라서, 예를 들면, 당해 라이브러리의 각각의 개별적인 멤버는 라이브러리의 모든 다른 멤버와 동일하거나 실질적으로 동일한 단백질-결합 세그먼트의 뉴클레오타이드 서열을 포함할 수 있고, 라이브러리의 모든 다른 멤버와 동일하거나 실질적으로 동일한 전사 종결 세그먼트의 뉴클레오타이드 서열을 포함할 수 있지만, DNA-표적화 RNA의 DNA 표적화 세그먼트의 뉴클레오타이드 서열 중의 라이브러리의 다른 멤버와 상이하다. 이러한 방식으로, 라이브러리는 상이한 표적 핵산에 결합하는 멤버를 포함할 수 있다.
유용성
본 명세서에 따르는 전사 조절 방법은 다양한 적용에서 용도를 찾고, 이도 또한 제공된다. 적용은 연구 적용, 진단 적용, 산업적 적용 및 치료 적용을 포함한다.
연구 적용은, 예를 들면, 표적 핵산의 전사를 감소시키거나 증가시키는 효과를, 예를 들면, 다운스트림 유전자의 발생, 대사, 발현 등에 대해 결정함을 포함한다.
고처리량 게놈 분석은 당해 전사 조절 방법을 사용하여 수행할 수 있고, 여기서 DNA-표적화 RNA의 DNA-표적화 세그먼트 만이 변화될 필요가 있는 반면, 단백질 결합 세그먼트 및 전사 종결 세그먼트는 (일부 경우에서) 일정하게 유지될 수 있다. 게놈 분석에 사용된 복수의 핵산을 포함하는 라이브러리(예: 당해 라이브러리)는 DNA-표적화 RNA-인코딩 뉴클레오타이드 서열에 작동가능하게 연결된 프로모터를 포함할 것이고, 여기서 각각의 핵산은 상이한 DNA-표적화 세그먼트, 공통의 단백질 결합 세그먼트 및 공통의 전사 종결 세그먼트를 포함할 것이다. 칩은 5 x 104 이상의 독특한 DNA-표적화 RNA를 함유할 수 있다. 적용은 대규모 표현형 검사, 유전자-대-기능 맵핑 및 메타-게놈 분석을 포함할 것이다.
본원에 개시된 당해 방법은 대사 공학 분야에서 용도를 찾는다. 전사 수준이 본원에서 개시된 바와 같이 적절한 DNA-표적화 RNA를 설계함으로써 효율적이고 예측가능하게 조절될 수 있기 때문에, 대사 경로(예: 생합성 경로)의 활성은 관심 대사 경로 내에서 특이적 효소의 수준을 조절함으로써(예: 증가되거나 감소된 전사를 통해) 정확하게 조절되고 조정될 수 있다. 관심 대사 경로는 화학물질(정밀한 화학물질, 연료, 항생제, 독소, 작용제, 길항제 등) 및/또는 약물 생산에 사용된 것들을 포함한다.
관심 생합성 경로는 (1) 메발론화 경로(예: HMG-CoA 리덕타제 경로)(아세틸-CoA를 테르페노이드/이소프레노이드를 포함하는 다양한 생체분자의 생합성용으로 사용되는 디메틸알릴 피로포스페이트(DMAPP) 및 이소펜테닐 피로포스페이트(IPP)로 전환시킨다), (2) 비-메발론화 경로(즉, "2-C-메틸-D-에리트리톨 4-포스페이트/1-데옥시-D-크실룰로즈 5-포스페이트 경로" 또는 "MEP/DOXP 경로" 또는 "DXP 경로"(메발론화 경로에 대한 대안적인 경로를 통해 피루베이트 및 글리세르알데하이드 3-포스페이트를 DMAPP 및 IPP로 전환시키는 대신, 또한 DMAPP 및 IPP를 생산한다), (3) 폴리케타이드 합성 경로(다양한 폴리케타이드 신타제 효소를 통해 다양한 폴리케타이드를 생산한다. 폴리케타이드는 화학요법에 사용된 자연 발생 소분자(예: 테트라사이클린 및 매크롤라이드)를 포함하고, 산업적으로 중요한 폴리케타이드는 라파마이신(면역억제제), 에리트로마이신(항생제), 로바스타틴(항콜레스테롤 약물) 및 에포틸론 B(항암 약물)을 포함한다), (4) 지방산 합성 경로, (5) DAHP(3-데옥시-D-아라비노-헵툴로소네이트 7-포스페이트) 합성 경로, (6) 잠재적인 바이오 연료(예: 단쇄 알콜 및 알칸, 지방산 메틸 에스테르 및 지방 알콜, 이소프레노이드 등)를 생산하는 경로 등을 포함하지만, 이에 국한되지 않는다.
네트워크 및 캐스케이드
본원에 개시된 방법은 제어의 통합된 네트워크(즉, 캐스케이드 또는 캐스케이드들)를 설계하기 위해 사용될 수 있다. 예를 들면, 당해 DNA-표적화 RNA/변이체 Cas9 부위 지향적 폴리펩타이드가 또 하나의 DNA-표적화 RNA 또는 또 하나의 당해 변이체 Cas9 부위 지향적 폴리펩타이드의 발현을 제어하기 위해(즉, 조절, 예를 들면, 증가시키고, 감소시키기 위해) 사용될 수 있다. 예를 들면, 제 1 DNA-표적화 RNA는 제 1 변이체 Cas9 부위 지향적 폴리펩타이드와 상이한 기능(예: 메틸트랜스퍼라제 활성, 데메틸라제 활성, 아세틸트랜스퍼라제 활성, 데아세틸라제 활성 등)을 갖는 제 2 키메라 dCas9 폴리펩타이드의 전사의 조절을 표적화하도록 설계될 수 있다. 또한, 상이한 dCas9 단백질(예: 상이한 종으로부터 유도됨)이 상이한 Cas9 핸들(즉, 단백질 결합 세그먼트)을 필요로 할 수 있기 때문에, 제 2 키메라 dCas9 폴리펩타이드는 상기 제 1 dCas9 폴리펩타이드와 상이한 종으로부터 유도될 수 있다. 따라서, 일부 경우에서, 제 2 키메라 dCas9 폴리펩타이드는 제 1 DNA-표적화 RNA와 상호작용할 수 없도록 선택될 수 있다. 다른 경우에서, 제 2 키메라 dCas9 폴리펩타이드는 제 1 DNA-표적화 RNA와 상호작용하도록 선택될 수 있다. 일부 이러한 경우에서, 두 개(또는 그 이상)의 dCas9 단백질의 활성은 경쟁할 수 있고(예를 들면, 폴리펩타이드가 반대 활성을 가질 경우) 또는 상승작용할 수 있다(예를 들면, 폴리펩타이드가 유사하거나 상승작용적 활성을 가질 경우). 마찬가지로, 상기에서 언급된 바와 같이, 네트워크 중의 임의의 복합체(즉, DNA-표적화 RNA/dCas9 폴리펩타이드)는 기타 DNA-표적화 RNA 또는 dCas9 폴리펩타이드를 제어하도록 설계될 수 있다. 당해 DNA-표적화 RNA 및 당해 변이체 Cas9 부위 지향적 폴리펩타이드는 임의의 목적하는 DNA 서열에서 표적화될 수 있기 때문에, 본원에 개시된 방법은 임의의 목적하는 표적의 발현을 제어하고 조절하기 위해 사용될 수 있다. 설계될 수 있는 통합 네트워크(즉, 상호작용의 캐스케이드)는 매우 단순한 것에서 매우 복합한 것에 이르고, 제한이 없다.
둘 이상의 성분(예: DNA-표적화 RNA, 활성제-RNA, 타겟터-RNA 또는 dCas9 폴리펩타이드)이 각각 또 하나의 DNA-표적화 RNA/dCas9 폴리펩타이드 복합체의 조절 제어하에 존재하는 네트워크에서, 네트워크의 한 성분의 발현 수준은 네트워크의 또 하나의 성분의 발현 수준에 영향을 미칠 수 있다(예: 발현을 증가시키거나 감소시킬 수 있다). 이 기전을 통해, 한 성분의 발현은 동일 네트워크 중의 상이한 성분의 발현에 영향을 미칠 수 있고, 네트워크는 다른 성분의 발현을 증가시키는 성분 뿐만 아니라 다른 성분의 발현을 감소시키는 성분의 혼합물을 포함할 수 있다. 당해 기술 분야의 숙련가에 의해 용이하게 이해되는 바와 같이, 한 성분의 발현 수준이 하나 이상의 상이한 성분(들)의 발현 수준에 영향을 미칠 수 있는 상기 예들은 설명하기 위한 것이고 제한하는 것은 아니다. 복합성의 추가 층이, 하나 이상의 성분이 조작가능하도록(즉, 실험적 제어하, 예를 들면, 온도 제어; 약물 제어, 즉 약물 유도성 제어; 광 제어 등) 변형될 경우(상기 기재된 바와 같이) 네트워크에 임의로 도입될 수 있다.
하나의 비제한적인 예로서, 제 1 DNA-표적화 RNA는 표적 치료/대사 유전자의 발현을 제어하는 제 2 DNA-표적화 RNA의 프로모터에 결합할 수 있다. 이러한 경우에, 제 1 DNA-표적화 RNA의 조건부 발현이 치료/대사 유전자를 간접적으로 활성화시킨다. 이 유형의 RNA 캐스케이드는, 예를 들면, 억제인자를 활성제로 용이하게 전환시키기에 유용하고, 표적 유전자의 발현의 논리학 또는 동력학을 제어하기 위해 사용될 수 있다.
당해 전사 조절 방법은 또한 약물 발견 및 표적 검증을 위해 사용될 수 있다.
키트
본 명세서는 당해 방법을 수행하기 위한 키트를 제공한다. 당해 키트는 a) 본 명세서의 DNA-표적화 RNA 또는 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산 및 b) 완충제를 포함하고, 여기서 DNA-표적화 RNA는 i) 표적 DNA 중의 표적 서열에 상보적인 뉴클레오타이드 서열을 포함하는 제 1 세그먼트; ii) 부위 지향적 폴리펩타이드와 상호작용하는 제 2 세그먼트; 및 iii) 전사 종결자를 포함한다. 일부 경우에서, DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산은 야생형 Cas9에 비해 감소된 엔도데옥시리보뉴클레아제 활성을 나타내는 변이체 Cas9 부위 지향적 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 추가로 포함한다.
일부 경우에서, 당해 키트는 야생형 Cas9에 비해 감소된 엔도데옥시리보뉴클레아제 활성을 나타내는 변이체 Cas9 부위 지향적 폴리펩타이드를 추가로 포함한다.
일부 경우에서, 당해 키트는 야생형 Cas9에 비해 감소된 엔도데옥시리보뉴클레아제 활성을 나타내는 변이체 Cas9 부위 지향적 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산을 추가로 포함한다.
대상체는 하나 이상의 추가의 시약을 추가로 포함할 수 있고, 여기서 이러한 추가의 시약은 완충제; 세척 완충제; 제어 시약; 제어 발현 벡터 또는 RNA 폴리뉴클레오타이드; DNA로부터 변이체 Cas9 부위 지향적 폴리펩타이드의 시험관내 생산을 위한 시약 등으로부터 선택될 수 있다. 일부 경우에서, 당해 키트에 포함된 변이체 Cas9 부위 지향적 폴리펩타이드는 상기 기재된 바와 같이, 융합 변이체 Cas9 부위 지향적 폴리펩타이드이다.
당해 키트의 성분은 별도의 용기에 존재할 수 있거나, 단일 용기에서 결합시킬 있다.
상기한 성분 이외에, 당해 키트는 당해 방법을 실시하기 위해 키트의 성분을 사용하기 위한 지침을 추가로 포함할 수 있다. 당해 방법을 실시하기 위한 지침은 일반적으로 적당한 기록 매체에 기록된다. 예를 들면, 지침은 종이 또는 플라스틱 등과 같은 기질 위에 인쇄될 수 있다. 이와 같이, 지침은 키트에 키트의 용기의 라벨링에서 포장 삽입물 또는 이의 성분(즉, 패키징 또는 서브패키징과 연관된) 등으로서 제공될 수 있다. 다른 구현예에서, 지침은 적당한 컴퓨터 판독가능한 저장 매체, 예를 들면, CD-ROM, 디스켓, 플래시 드라이브 등에 존재하는 전자 저장 데이터 파일로서 존재한다. 다른 구현예에서, 실제 지침은 키트에 존재하지 않고, 원격 공급원으로부터, 예를 들면, 인터넷을 통해 지침을 수득하는 수단이 제공된다. 이 구현예의 예는 지침이 검토될 수 있고/있거나 지침이 다운로드될 수 있는 웹 주소를 포함하는 키트이다. 지침을 갖는 경우와 같이, 지침을 수득하기 위한 이 수단은 적합한 기질 상에 기록된다.
실시예
하기 실시예들은 당해 기술 분야의 숙련가에게 본 발명을 제조하고 사용하는 방법에 대한 완전한 명세서 및 설명을 제공하기 위해 기재되어 있고, 본 발명자들이 그들의 발명으로서 간주하는 범위를 제한하려는 것이 아니고, 그들은 이하 실험이 수행되는 모든 실험이거나 유일한 실험임을 나타내도록 의도된 것도 아니다. 사용된 수(예: 양, 온도 등)에 대하여 정확도를 보장하기 위해 노력이 이루어졌지만, 약간의 실험적 오차 및 편차는 설명되어야 한다. 달리 지시되지 않으면, 부는 중량부이고, 분자량은 중량평균분자량이고, 온도는 섭씨온도이고, 압력은 대기압 또는 거의 대기압이다. 표준 약어, 예를 들어, bp, 염기쌍(들); kb, 킬로염기(들); pl, 피코리터(들); s 또는 sec, 초(들); min, 분(들); h 또는 hr, 시간(들); aa, 아미노산(들); kb, 킬로염기(들); bp, 염기쌍(들); nt, 뉴클레오타이드(들); i.m., 근육내(로); i.p., 복강내(로); s.c., 피하(로) 등이 사용될 수 있다.
실시예 1: 표적 DNA를 변형시키기 위한 Cas9의 사용
물질 및 방법
박테리아 균주 및 배양 조건
THY 배지(0.2% 효모 추출물(옥소이드)이 보충된 토드 휴이트 액체배지(THB, 박토, 벡토 디킨슨)) 중에서 또는 3% 양 혈액이 보충된 TSA(트립티카제 대두 한천, BBL, 벡톤 디킨슨) 상에서 배양된 스트렙토코쿠스 파이오제네스를 진탕 없이 5% CO2가 보충된 대기에서 37℃에서 배양시켰다. 루리아-베르타니(LB) 배지 및 한천 중에서 배양된 에스케리치아 콜리를 진탕하면서 37℃에서 배양시켰다. 필요한 경우에, 적당한 항생제를 다음 최종 농도로 배지에 첨가했다: 이. 콜라이의 경우, 암피실린 100㎍/ml; 에스케리치아 콜리의 경우, 클로르암페니콜 33㎍/ml; 이. 콜라이의 경우, 카나마이신 25㎍/ml 및 에스. 파이오제네스의 경우, 300㎍/ml. 박테리아 세포 성장은 마이크로플레이트 판독기(SLT 스펙트럼 판독기)를 사용하여 620nm에서 배양 분취량의 광학 밀도를 측정하여 주기적으로 모니터링했다.
박테리아 세포의 형질전환
이. 콜라이 세포로 플라스미드 DNA 형질전환은 표준 열 충격 프로토콜에 따라 수행하였다. 에스. 파이오제네스의 형질전환은 일부 변형으로 이전에 기재된 바와 같이 수행하였다. 플라스미드 유지에 대한 생체내 CRISPR/CAS 활성을 모니터하기 위해 수행된 형질전환 검정은 이미 기술된 바와 같이 본질적으로 수행하였다. 간단하게, 에스. 파이오제네스의 전기반응능 세포를 동일한 세포 밀도로 균등화시켰고, 500ng의 플라스미드 DNA로 전기천공시켰다. 모든 형질전환은 2 내지 3회 플레이팅하고, 실험을 통계 분석을 위해 반응능 세포의 상이한 배치로 독립적으로 3회 수행하였다. 형질전환 효율성은 DNA 1㎍당 CFU(콜로니 형성 단위)로서 계산하였다. 대조군 형질전환은 멸균수 및 골격 벡터 pEC85로 수행하였다.
DNA 조작
DNA 제조, 증폭, 소화, 결찰, 정제, 아가로스 겔 전기영동을 포함하는 DNA 조작은 최소 변형으로 표준 기술에 따라 수행하였다. 시험관내 절단 및 에스. 파이오제네스 형질전환 검정을 위한 프로토스페이서 플라스미드를 이미 기술된 바와(4) 같이 구성했다. 시험관 절단 검정을 위한 추가의 pUC19-기반 프로토스페이서 플라스미드는 pUC19 중의 소화된 EcoRI 및 BanHI 부위 사이에 어닐링된 올리고뉴클레오타이드를 결찰시켜 생성하였다. GFP 유전자 함유 플라스미드는 이전에 기술되었다(41). 키트(Qiagen)를 DNA 정제 및 플라스미드 제조용으로 사용하였다. 플라스미드 돌연변이유발은 QuikChangeR II XL 키트(Stratagene) 또는 QuikChange 부위 지향적 돌연변이유발 키트(Agilent)를 사용하여 수행하였다. VBC-바이오테크 서비시즈, 시그마-알드리치 및 인테크레이티드 DNA 테크롤로지스는 합성 올리고뉴클레오타이드 및 RNA를 공급한다.
시험관내 전사 템플레이트를 위한 올리고뉴클레오타이드
에스. 파이오제네스의 시험관내 전사된 CRISPR 유형 II-A tracrRNA 및 crRNA를 위한 (chr. DNA SF370 상의 tracrRNA-PCR을 위한; 두 올리고뉴클레오타이드의 crRNA-어닐링을 위한) 템플레이트
T7-tracrRNA (75nt)
OLEC1521 (F 5' tracrRNA): 서열번호 340
OLEC1522 (R 3' tracrRNA): 서열번호 341
T7-crRNA (템플레이트)
OLEC2176 (F crRNA-sp1): 서열번호 342
OLEC2178 (R crRNA-sp1): 서열번호 343
OLEC2177 (F crRNA-sp2): 서열번호 344
OLEC2179 (R crRNA-sp2): 서열번호 345
시험관내 전사된 엔. 메닌자이티디스 tracrRNA 및 조작된 crRNA - sp2 를 위한 (chr. DNA Z2491 상의 tracrRNA - PCR 을 위한; 두 올리고뉴클레오타이드의 crRNA -어닐링을 위한) 템플레이트
T7-tracrRNA
OLEC2205 (F 예상된 5'): 서열번호 346
OLEC2206 (R 예상된 3'): 서열번호 347
T7-crRNA (템플레이트)
OLEC2209 (F sp2(speM) + N.m. 반복): 서열번호 348
OLEC2214 (R sp2(speM) + N.m. 반복): 서열번호 349
시험관내 전사된 엘. 이노쿠아 tracrRNA 및 조작된 crRNA - sp2 를 위한 ( chr . DNA Clip11262 상의 tracrRNA - PCR 을 위한; 두 올리고뉴클레오타이드의 crRNA - 어닐 링을 위한) 템플레이트
T7-tracrRNA
OLEC2203 (F 예상된 5'): 서열번호 350
OLEC2204 (R 예상된 3'): 서열번호 351
T7-crRNA (템플레이트)
OLEC2207 (F sp2(speM) + L.in. 반복): 서열번호 352
OLEC2212 (R sp2(speM) + L.in. 반복): 서열번호 353
시험관내 및 생체내 연구를 위한 프로토스페이서를 갖는 플라스미드를 작제하기 위한 올리고뉴클레오타이드
시험관내 및 에스. 파이오제네스의 speM (스페이서 2 (CRISPR 유형 II-A, SF370; MGAS8232로부터 프로토스페이서 프로파아지 Ø8232.3) 분석을 위한 플라스미드(템플레이트: chr. DNA MGAS8232 또는 speM 단편을 함유하는 플라스미드)
pEC287
OLEC1555 (F speM): 서열번호 354
OLEC1556 (R speM): 서열번호 355
pEC488
OLEC2145 (F speM): 서열번호 356
OLEC2146 (R speM): 서열번호 357
pEC370
OLEC1593 (F pEC488 프로토스페이서 2 A22G): 서열번호 358
OLEC1594 (R pEC488 프로토스페이서 2 A22G): 서열번호 359
pEC371
OLEC1595 (F pEC488 프로토스페이서 2 T10C): 서열번호 360
OLEC1596 (R pEC488 프로토스페이서 2 T10C): 서열번호 361
pEC372
OLEC2185 (F pEC488 프로토스페이서 2 T7A): 서열번호 362
OLEC2186 (R pEC488 프로토스페이서 2 T7A): 서열번호 363
pEC373
OLEC2187 (F pEC488 프로토스페이서 2 A6T): 서열번호 364
OLEC2188 (R pEC488 프로토스페이서 2 A6T): 서열번호 365
pEC374
OLEC2235 (F pEC488 프로토스페이서 2 A5T): 서열번호 366
OLEC2236 (R pEC488 프로토스페이서 2 A5T): 서열번호 367
pEC375
OLEC2233 (F pEC488 프로토스페이서 2 A4T): 서열번호 368
OLEC2234 (R pEC488 프로토스페이서 2 A4T): 서열번호 369
pEC376
OLEC2189 (F pEC488 프로토스페이서 2 A3T): 서열번호 370
OLEC2190 (R pEC488 프로토스페이서 2 A3T): 서열번호 371
pEC377
OLEC2191 (F pEC488 프로토스페이서 2 PAM G1C): 서열번호 372
OLEC2192 (R pEC488 프로토스페이서 2 PAM G1C): 서열번호 373
pEC378
OLEC2237 (F pEC488 프로토스페이서 2 PAM GG1, 2CC): 서열번호 374
OLEC2238 (R pEC488 프로토스페이서 2 PAM GG1, 2CC): 서열번호 375
시험관내 및 에스 . 파이오제네스에서 SPy _0700 ( 스페이서 1 ( CRISPR 유형 II-A, SF370 ; SF370 으로부터 프로토스페이서 프로파아지 Ø370.1) 분석을 위한 플라스미드(템플레이트: chr. DNA SF370 또는 SPy_0700 단편을 함유하는 플라스미드)
pEC489
OLEC2106 (F Spy_0700): 서열번호 376
OLEC2107 (R Spy_0700): 서열번호 377
pEC573
OLEC2941 (F PAM TG1, 2GG): 서열번호 378
OLEC2942 (R PAM TG1, 2GG): 서열번호 379
서열분석에 의한 플라스미드 작제물 및 절단 부위를 입증하기 위한 올리고뉴클레오타이드
ColE1 (pEC85)
oliRN228 (R 서열분석): 서열번호 380
speM (pEC287)
OLEC1557 (F 서열분석): 서열번호 381
OLEC1556 (R 서열분석): 서열번호 382
repDEG-pAMbeta1 (pEC85)
OLEC787 (F 서열분석): 서열번호 383
시험관내 절단 검정을 위한 올리고뉴클레오타이드
crRNA
스페이서 1 crRNA (1-42): 서열번호 384
스페이서 2 crRNA (1-42): 서열번호 385
스페이서 4 crRNA (1-42): 서열번호 386
스페이서 2 crRNA (1-36): 서열번호 387
스페이서 2 crRNA (1-32): 서열번호 388
스페이서 2 crRNA (11-42): 서열번호 389
tracrRNA
(4-89): 서열번호 390
(15-89): 서열번호 391
(23-89): 서열번호 392
(15-53): 서열번호 393
(15-44): 서열번호 394
(15-36): 서열번호 395
(23-53): 서열번호 396
(23-48): 서열번호 397
(23-44): 서열번호 398
(1-26): 서열번호 399
키메라 RNA
스페이서 1 - 키메라 A: 서열번호 400
스페이서 1 - 키메라 B: 서열번호 401
스페이서 2 - 키메라 A: 서열번호 402
스페이서 2 - 키메라 B: 서열번호 403
스페이서 4 - 키메라 A: 서열번호 404
스페이서 4 - 키메라 B: 서열번호 405
GFP1: 서열번호 406
GFP2: 서열번호 407
GFP3: 서열번호 408
GFP4: 서열번호 409
GFP5: 서열번호 410
절단 검정용 기질로서 DNA 올리고뉴클레오타이드 (프로토스페이서 굵게, PAM 밑줄)
프로토스페이서 1 - 상보적 - WT: 서열번호 411
프로토스페이서 1 - 비상보적 - WT: 서열번호 412
프로토스페이서 2 - 상보적 - WT: 서열번호 413
프로토스페이서 2 - 비상보적 - WT: 서열번호 414
프로토스페이서 4 - 상보적 - WT: 서열번호 415
프로토스페이서 4 - 비상보적 - WT: 서열번호 416
프로토스페이서 2 - 상보적 - PAM1: 서열번호 417
프로토스페이서 2 - 비상보적 - PAM1: 서열번호 418
프로토스페이서 2 - 상보적 - PAM2: 서열번호 419
프로토스페이서 2 - 비상보적 - PAM2: 서열번호 420
프로토스페이서 4 - 상보적 - PAM1: 서열번호 421
프로토스페이서 4 - 비상보적 - PAM1: 서열번호 422
프로토스페이서 4 - 상보적 - PAM2: 서열번호 423
프로토스페이서 4 - 비상보적 - PAM2: 서열번호 424
RNA 의 시험관내 전사 및 정제
RNA는 T7 플래시 시험관내 전사 키트(Epicentre, Illumina company) 및 T7 프로모터 서열을 수반하는 PCR-생성된 DNA 템플레이트를 사용하여 시험관내 전사시켰다. RNA는 겔 정제시키고, 사용 직전에 품질 체크하였다. 에스. 파이오제네스 SF370, 리스테리아 이노쿠아 Clip 11262 및 나이세리아 메닌자이티디스 A Z2491로부터 RNA 템플레이트의 제조용으로 사용된 프라이머는 상기 기재된다.
단백질 정제
Cas9를 인코딩하는 서열(잔기 1-1368)은 에스. 파이오제네스 SF370의 게놈 DNA로부터 PCR증폭시키고, 결찰 독립적 클로닝(LIC)을 사용하여 커스텀 pET-기반 발현 벡터에 삽입하였다. 수득한 융합 작제물은 N-말단 헥사히스티딘-말토오스 결합 단백질(His6-MBP) 태그에 이어, 담배 부식 작용 바이러스(TEV) 프토테아제 절단 부위를 함유하는 펩타이드 서열을 함유했다. 단백질은 이. 콜라이 균주 BL21 로제타 2(DE3)(EMD Biosciences)에서 발현시키고, 0.2mM IPTG로 유도한 다음 18℃에서 16시간 동안 2xTY 배지에서 성장시켰다. 단백질은 친화성, 이온 교환 및 크기 배제 크로마토그래피 단계의 조합으로 정제시켰다. 간단하게, 세포를 균질기(Avestin) 중의 20 mM Tris pH 8.0, 500 mM NaCl, 1 mM TCEP (프토테아제 억제제 칵테일(Roche)이 보충됨)에 용해시켰다. 정화된 용해물을 배치로 Ni-NTA 아가로스(Qiagen)에 결합시켰다. 수지를 20 mM Tris pH 8.0, 500 mM NaCl로 광범위하게 세척하고, 결합된 단백질을 20 mM Tris pH 8.0, 250 mM NaCl, 10% 글리세롤에서 용출시켰다. His6-MBP 친화성 태그를 TEV 프로테아제로 절단하여 제거하고, 단백질을 밤새 20 mM HEPES pH 7.5, 150 mM KCl, 1 mM TCEP, 10% 글리세롤에 대해 투석했다. 절단된 Cas9 단백질은 100mM-1M KCl의 선형 구배로 용출시키면서 5ml SP 세파로스 HiTrap 컬럼(GE Life Sciences) 상에서 정제로 융합 태그로부터 분리시켰다. 단백질은 20 mM HEPES pH 7.5, 150 mM KCl 및 1 mM TCEP 중의 슈퍼덱스 200 16/60 컬럼 상에서 크기 배제 크로마토그래피로 추가로 정제했다. 용출된 단백질을 약 8mg/ml로 농축시키고, 액체 질소로 플래시 동결시키고, -80℃에서 저장하였다. Cas9 D10A, H840A 및 D10A/H840A 포인트 돌연변이체는 QuikChange 부위 지향적 돌연변이유발 키트(Agilent)를 사용하여 생성하였고, DNA 서열 분석으로 확인했다. 단백질은 야생형 Cas9 단백질의 경우와 동일한 절차에 따라 정제시켰다.
스트렙토코쿠스 서모필러스 (LMD-9,YP_820832.1), 엘. 이노쿠아 (Clip11262, NP_472073.1), 캠필로박터 제주니 (subsp. 제주니 NCTC 11168, YP_002344900.1) 및 엔. 메닌자이티디스 (Z2491, YP_002342100.1)로부터 Cas9 오쏘로그를 BL21 로제타 (DE3) pLysS 세포(Novagen)에서 His6-MBP (엔. 메닌자이티디스 및 씨. 제주니), His6-티오레독신 (엘. 이노쿠아) 및 His6-GST (에스. 서모필러스) 융합 단백질로서 발현시키고, 본질적으로 하기 변형과 함께 에스. 파이오제네스 Cas9의 경우와 같이 정제시켰다. 다량의 공동-정제 핵산에 기인하여, 4개의 Cas9 단백질 모두를 겔 여과 전에 결합된 단백질을 100mM 내지 2M KCl의 선형 구배로 용출시키면서 추가의 헤파인 세파로스 단계에 의해 정제시켰다. 이는 씨. 제주니, 엔. 메닌자이티디스 및 엘. 이노쿠아 단백질로부터 핵산 오염을 성공적으로 제거하였지만, 에스. 서모필러스 Cas9 제조로부터 공동-정제 핵산을 제거하지는 못했다. 모든 단백질을 20 mM HEPES pH 7.5, 150 mM KCl 및 1 mM TCEP 중에서 1 내지 8mg/ml로 농축시키고, 액체 N2로 플래시 동결시키고, -80℃에서 저장하였다.
플라스미드 DNA 절단 검정
합성 또는 시험관내-전사된 tracrRNA 및 crRNA를 95℃로 가열하여 실온으로 서서히 냉각시킴으로써 반응 전에 전어닐링시켰다. 천연 또는 제한 소화-선형화된 플라스미드 DNA(300ng(약 8nM))를 10 mM MgCl2를 사용하거나 사용하지 않고 Cas9 플라스미드 절단 완충제(20 mM HEPES pH 7.5, 150 mM KCl, 0.5 mM DTT, 0.1 mM EDTA)에서 정제된 Cas9 단백질(50 내지 500nM) 및 tracrRNA:crRNA 듀플렉스(50 내지 500 nM, 1:1)로 37℃에서 60분 동안 배양하였다. 반응을 250mN EDTA를 함유하는 5X DNA 부하 완충제로 정지시킨 다음, 0.8 또는 1% 아가로스 겔 전기영동으로 분해시키고, 브롬화에티듐 염색으로 가시화하였다. Cas9 돌연변이체 절단 검정을 위해, 반응을 아가로스 겔 상에 부하 전에 5X SDS 부하 완충제(30% 글리세롤, 1.2% SDS, 250mM EDTA)로 정지시켰다.
금속-의존 절단 검정
프로토스페이서 2 플라스미드 DNA(5nM)를 1, 5 또는 10 mM MgCl2, 1 또는 10 mM의 MnCl2, CaCl2, ZnCl2, CoCl2, NiSO4 또는 CuSO4가 보충된 절단 완충제 (20 mM HEPES pH 7.5, 150 mM KCl, 0.5 mM DTT, 0.1 mM EDTA) 중에서 50 nM tracrRNA:crRNA-sp2로 예비배양된 Cas9(50nM)로 37℃에서 1시간 동안 배양하였다. 반응은 5X SDS 부하 완충제(30% 글리세롤, 1.2% SDS, 250mM EDTA)를 첨가하여 정지시키고, 1% 아가로스 겔 전기영동으로 분해시키고, 브롬화에티듐 염색으로 가시화하였다.
단일-턴오버 검정
Cas9(25nM)를 절단 완충제(20 mM HEPES pH 7.5, 150 mM KCl, 10 mM MgCl2, 0.5 mM DTT, 0.1 mM EDTA)에서 듀플렉스화 tracrRNA:crRNA-sp2 (25 nM, 1:1) 또는 전어닐링되지 않은 두 RNA(25 nM)로 37℃에서 15분 동안 예비배양하고, 반응은 프로토스페이서 2 플라스미드 DNA(5nM)를 첨가하여 개시하였다. 반응 혼합물을 37℃에서 배양하였다. 소정의 시간 간격으로, 샘플을 반응물로부터 회수하고, 5X SDS 부하 완충제(30% 글리세롤, 1.2% SDS, 250mM EDTA)를 첨가하여 반응을 정지시키고, 1% 아가로스 겔 전기영동 및 브롬화에티듐 염색으로 절단을 모니터링하였다. Cas9 및 RNA의 예비배양 없이 단일 턴오버 동역학에 대해 동일하게 수행하였고, 여기서 프로토스페이서 2 플라스미드 DNA(5nM)를 절단 완충제에서 듀플렉스 tracrRNA:crRNA-sp2 (25 nM) 또는 전어닐링되지 않은 두 RNA(25 nM)와 혼합하고, 반응은 Cas9(25nM)를 첨가하여 개시하였다. 절단율은 농도학으로 분석하였고, 3개의 독립적인 실험의 평균을 시간에 대해 플롯팅하였다. 데이터는 비선형 회귀 분석에 의해 적합화시켰고, 절단 속도(kobs[min-1])를 계산하였다.
다중-턴오버 검정
Cas9(1nM)를 절단 완충제(20 mM HEPES pH 7.5, 150 mM KCl, 10 mM MgCl2, 0.5 mM DTT, 0.1 mM EDTA)에서 전-어닐링된 tracrRNA:crRNA-sp2(1nM, 1:1)로 37℃에서 15분 동안 예비배양하였다. 반응은 프로토스페이서 2 플라스미드 DNA(5nM)를 첨가하여 개시하였다. 소정의 시간 간격으로, 샘플을 회수하고, 5X SDS 부하 완충제(30% 글리세롤, 1.2% SDS, 250mM EDTA)를 첨가하여 반응을 정지시켰다. 절단 반응물을 1% 아가로스 겔 전기영동으로 분해시키고, 브롬화에티듐으로 염색시키고, 절단율은 농도학으로 분석하였다. 4개의 독립적인 실험의 결과를 시간(분)에 대해 플롯팅하였다.
올리고뉴클레오타이드 DNA 절단 검정
DNA 올리고뉴클레오타이드(10 pmol)를 50μL 반응물 중에서 37℃에서 30분 동안 1X T4 폴리뉴클레오타이드 키나제 반응 완충제 중의 5 유닛 T4 폴리뉴클레오타이드 키나제(New England Biolabs) 및 약 3 내지 6 pmol(약 20 내지 40mCi)[γ-32P]-ATP(Promega)로 배양하여 방사선표지화하였다. 열 불활성화 후(65℃에서 20분 동안), 반응물을 일루스트라 마이크로스핀 G-25 컬럼(GE Healthcare)을 통해 정제하여 비편입된 라벨을 제거하였다. 듀플렉스 기질(100nM)은 표지된 올리고뉴클레오타이드를 등몰량의 비표지된 상보적 올리고뉴클레오타이드로 95℃에서 3분 동안 어닐링한 후, 실온으로 서서히 냉각시켜 생성하였다. 절단 검정을 위해, tracrRNA 및 crRNA를 95℃로 30초 동안 가열한 다음, 실온으로 서서히 냉각시켜 어닐링시켰다. Cas9(500nM 최종 농도)를 총 용적 9㎕로 절단 검정 완충제(20 mM HEPES pH 7.5, 100 mM KCl, 5 mM MgCl2, 1 mM DTT, 5% 글리세롤) 중에서 어닐링된 tracrRNA:crRNA 듀플렉스(500 nM)로 예비배양했다. 반응을 1㎕ 표적 DNA(10nM)를 첨가하여 개시하고, 1시간 동안 37℃에서 배양하였다. 반응물을 20㎕의 부하 염료(5 mM EDTA, 0.025% SDS, 포름아미드 중의 5% 글리세롤)를 첨가하여 급냉시키고, 95℃로 5분 동안 가열하였다. 절단 생성물을 7M 우레아를 함유하는 12% 변성 폴리아크릴아미드 겔 상에서 분해하고, 포스포르이미지화(Storm, GE Life Sciences)로 가시화하였다. PAM 요건을 시험하는 절단 검정(도 13b)은 전-어닐링화되고, 8% 천연 아크릴아미드 겔 상에서 정제시킨 다음, 두 5' 말단에서 방사선표지된 DNA 듀플렉스 기질을 사용하여 수행하였다. 반응을 설정하고, 상기한 바와 같이 분석하였다.
전기영동 이동도 변화 검정
표적 DNA 듀플렉스는 탈이온수 중의 각각의 가닥(10nmol)을 혼합하고, 95℃로 3분 동안 가열하고, 실온으로 서서히 냉각시켜 형성하였다. 모든 DNA는 1X TBE를 함유하는 8% 천연 겔 상에서 정제시켰다. DNA 밴드는 UV 셰도잉에 의해 가시화하고, 절개하고, 겔 조각을 DEPC 처리된 H2O에 침지시켜 용출시켰다. 용출된 DNA를 에탄올 침전시키고, DEPC 처리된 H2O에 용해시켰다. DNA 샘플을 37℃에서 30분 동안 T4 폴리뉴클레오타이드 키나제(New England Biolabs)를 사용하여 [γ-32P]-ATP로 5' 말단 표지화하였다. PNK를 65℃에서 20분 동안 열 변성시키고, 비편입된 방사선표지는 일루스트라 마이크로스핀 G-25 컬럼(GE Healthcare)을 사용하여 제거하였다. 결합 검정은 총 용적 10㎕로 20 mM HEPES pH 7.5, 100 mM KCl, 5 mM MgCl2, 1 mM DTT 및 10% 글리세롤을 함유하는 완충제에서 수행하였다. Cas9 D10A/H840A 이중 돌연변이체를 등몰량의 전어닐링된 tracrRNA:crRNA 듀플렉스로 프로그래밍하고, 100pM 내지 1μM로 적정하였다. 방사선표지된 DNA를 최종 농도 20pM으로 첨가하였다. 샘플을 1시간 동안 37℃에서 배양하고, 4℃에서 1X TBE 및 5mM MgCl2를 함유하는 8% 천연 폴리아크릴아미드 겔 상에서 분해시켰다. 겔을 건조시키고, DNA를 포스포르이미지화로 가시화하였다.
DNA 및 단백질 서열의 인 실리코 분석
벡터 NTI 패키지(Invitrogen)를 DNA 서열 분석(벡터 NTI) 및 단백질(AlignX)의 비교 서열 분석을 위해 사용하였다.
RNA 구조 및 공동- 폴딩의 인 실리코 모델링
인 실리코 예측은 비엔나 RNA 패키지 알고리즘(42, 43)을 사용하여 수행하였다. RNA 2차 구조 및 공동-폴딩 모델은 각각 RNA폴드 및 RNA공동폴드로 예측하였고, VARNA(44)로 가시화하였다.
결과
박테리아 및 고세균은 유기체를 바이러스 및 플라스미드(1-3) 침입으로부터 보호하는 군집형태의 규칙적으로 공간이 사이에 두어진 짧은 회귀성 반복체(CRISPR)/CRISPR 연관된(CAS)으로 칭명되는 RNA 매개된 적응 방어 시스템을 진화시켰다. 본 발명자들은 이러한 시스템의 서브세트에서, 트랜스-활성화 crRNA(tracrRNA)에 대한 염기 짝짓기된 성숙 crRNA는 표적 DNA에 이중-가닥(ds) 파괴를 도입하도록 CRISPR-연관된 단백질 Cas9에 지시하는 두-RNA 구조를 형성한다는 것을 보여준다. crRNA-가이드 서열에 상보적인 부위에서, Cas9 HNH 뉴클레아제 도메인은 상보적 가닥을 절단하는 반면, Cas9 RuvG-형 도메인은 비상보적 가닥을 절단한다. 이중-tracrRNA:crRNA는, 단일 RNA 키메라로서 조작될 경우, 또한 서열-특이적 Cas9 dsDNA 절단을 지시한다. 이러한 연구는 부위 특이적 DNA 절단을 위해 이중-RNA를 사용하는 엔도뉴클레아제의 패밀리를 나타내고, RNA-프로그래밍가능한 게놈 편집을 위해 상기 시스템을 활용하는 능력을 강조한다.
CRISPR/Cas 방어 시스템은 서열 특이적 검출 및 외래 핵산의 사일런싱을 위한 작은 RNA에 의존한다. CRISPR/Cas 시스템은 동일한 반복체(1-3)가 산재된 게놈-표적화 서열(스페이서로 칭명됨)로 구성된 오페론(들) 및 CRISPR 어레이(들)로 조직된 cas 유전자로 구성되어 있다. CRISPR/Cas-매개된 면역성은 3 단계로 일어난다. 적응 단계에서, 하나 이상의 CRISPR 유전자 좌를 보유하는 박테리아 및 고세균은 외래 서열의 짧은 단편(프로토스페이서)을 CRISPR 어레이(1-3)의 근위 말단에서 숙주 염색체에 통합함으로써 바이러스 또는 플라스미드 챌린지에 반응한다. 발현 및 간섭 단계에서, 효소 절단이 따르는 반복 스페이서 요소의 전구체 CRISPR RNA(전-crRNA) 분자로의 전사는 침입하는 바이러스 또는 플라스미드 표적(4-11)의 상보적 프로토스페이서 서열과 쌍을 이룰 수 있는 짧은 crRNA를 수득한다. crRNA에 의한 표적 인식은 crRNA(10, 12-20)와의 복합체에서 기능을 하는 Cas 단백질에 의한 외래 서열의 사일런싱을 지시한다.
3 유형의 CRISPR/Cas 시스템(21-23)이 존재한다. 유형 I 및 III 시스템은 일부 중요한 특징을 공유한다: 특화된 Cas 엔도뉴클레아제는 전-crRNA를 처리하고, 일단 성숙된 각각 crRNA는 crRNA에 대해 상보적인 핵산을 인식하고 절단할 수 있는 대규모 다중-Cas 단백질 복합체로 조립한다. 대조적으로, 유형 II 시스템은 전-crRNA 중의 반복 서열에 상보적인 트랜스-활성화 crRNA(tracrRNA)가 Cas9(이전에 Csn1) 단백질(도 15)(4,24)의 존재하에 이중-가닥(ds) RNA특이적 리보뉴클레아제 RNase III에 의한 처리를 유발하는 상이한 기전에 의해 전-crRNA를 처리한다. Cas9는 외래 DNA(25-27)의 crRNA-유도된 사일런싱을 초래하는 유일한 단백질인 것으로 간주된다.
본 발명자들은 유형 II 시스템에서, Cas9 단백질은 표적 dsDNA를 절단하기 위해 활성화 tracrRNA와 표적화 crRNA 사이에 형성된 염기 짝짓기된 구조를 필요로 하는 효소의 패밀리를 구성한다는 것을 보여준다. 부위 특이적 절단은 crRNA와 표적 프로토스페이서 DNA 사이에 염기-짝짓기 상보성 ? 표적 DNA 중의 상보적 영역에 병렬된 짧은 모티프[프로토스페이서 인접 모티프(PAM)로서 칭명됨] 둘 다에 의해 결정된 위치에서 일어난다. 본 연구는 Cas9 엔도뉴클레아제 패밀리가 특이적 DNA 부위를 절단하기 위해 단일 RNA 분자로 프로그래밍되어 게놈 표적화 및 편집을 위해 dsDNA 절단을 생성하기 위한 간단한 만능 RNA-유도된 시스템의 개발을 촉진시킬 수 있음을 추가로 입증한다.
Cas9 는 두 RNA 에 의해 유도된 DNA 엔도뉴클레아제이다
유형 II 시스템의 홀마크 단백질인 Cas9는 crRNA 성숙 및 crRNA-유도된 DNA 간섭(도 15)(4, 25-27) 둘 다에 연관되는 것으로 가정되어 있다. Cas9는 crRNA 성숙(4)에 연관되지만, 표적 DNA 파괴에 이의 직접적인 참여는 조사되지 않았다. Cas9가 표적 DNA 절단을 할 수 있는지의 여부 및 표적 DNA 절단을 하는 방법을 시험하기 위해, 본 발명자는 병원체 스트렙토코쿠스 파이오제네스(도 16, 상보적 물질 및 방법 참조)로부터 유도된 Cas9 단백질을 정제하기 위해 과발현 시스템을 사용하고, 성숙 crRNA에 상보적인 프로토스페이서 서열 및 진실한 PAM을 포함하는 올리고뉴클레오타이드 듀플렉스 또는 플라스미드 DNA를 절단하는 이의 능력을 시험했다. 본 발명자들은 성숙 crRNA 단독은 Cas9-촉매된 플라스미드 DNA 절단을 지시할 수 없었다(도 10a 및 도 17a는 것을 밝혀내었다. 그러나, crRNA의 반복 서열과 쌍을 이룰 수 있고 이 시스템에서 crRNA 성숙에 필수적인 tracrRNA의 첨가는 플라스미드DNA를 절단시키는 Cas9를 유발한다(도 10a 및 도 17a). 절단 반응은 마그네슘 및 DNA에 상보적인 crRNA 서열의 존재 둘 다를 필요로 하고; tracrRNA염기 짝짓기 할 수 있지만 비동족 표적 DNA-결합 서열을 함유하는 crRNA는 Cas9 촉매된 플라스미드 절단을 지지하지 않는다(도 10a; 도 17a, crRNA-sp2와 crRNA-sp1 비교; 및 도 18a). 본 발명자들은 짧은 선형 dsDNA 기질로 유사한 결과를 수득하였다(도 10b 및 도 17, b 및 c). 따라서, 트랜스-활성화 tracrRNA는 효소 RNase III(4)에 의한 전-crRNA 처리를 유발하고, 후속적으로 Cas9에 의해 crRNA-유도된 DNA 절단을 활성화하는 두 개의 중요한 기능을 갖는 작은 비코딩 RNA이다.
tracrRNA:crRNA-유도된 Cas9에 의한 플라스미드 및 짧은 선형 dsDNA 둘 다의 절단은 부위특이적이다(도 10, c 내지 e, 및 도 19, a 및 b). 플라스미드 DNA 절단은 PAM 서열의 3개의 염기쌍 업스트림 위치에서 무딘 말단을 생성했다(도 10, c 및 e, 및 도 19, a 및 c)(26). 유사하게, 짧은 dsDNA 듀플렉스 이내에서, crRNA 중의 표적 결합 서열에 상보적인 DNA 가닥(상보적 가닥)이 PAM의 3개의 염기쌍 업스트림 부위에서 절단된다(도 10, d 및 e, 및 도 19, b 및 c). 비상보적 DNA 가닥은 PAM의 3 내지 8개 염기쌍 업스트림 내의 하나 이상의 부위에서 절단된다. 추가의 조사는 비상보적 가닥이 먼저 엔도핵산분해로 절단된 다음, 3'-5' 엑소뉴클레아제 활성으로 조절됨을 나타냈다(도 18b). 단일-턴오버 조건하에 Cas9에 의한 절단속도는 제한 엔도뉴클레아제(도 20a)의 것들과 비교할만한 0.3 내지 1min-1의 범위인 반면, 5배 몰 과량의 기질 DNA에 의한 야생형(WT) Cas9-tracrRNA:crRNA 복합체의 배양은 이중-RNA-유도된 Cas9가 다중-턴오버 효소이다는 증거를 제공한다(도 20b). CRISPR 유형 I 캐스케이드 복합체(18)와 대조적으로, Cas9는 선형화된 및 초나선화 플라스미드 둘 다를 절단한다(도 10a 및 11a). 따라서, 침입성 플라스미드는, 원칙적으로 상이한 crRNA로 프로그래밍된 Cas9 단백질로 다수회 절단될 수 있다.
도 10 (A) Cas9는 75-뉴클레오타이드 tracrRNA의 존재 또는 부재하에 42-뉴클레오타이드 crRNA-sp2(스페이서 2 서열을 함유하는 crRNA)로 프로그래밍하였다. 복합체를 스페이서 2에 상보적인 서열 및 기능적 PAM을 포함하는 원형 또는 Xhol-선형화된 플라스미드 DNA에 부가하였다. crRNA-sp1, 특이성 대조군; M, DNA 마커; kbp, 킬로-염기쌍. 도 17a 참조. (B) Cas9는 crRNA-sp2 및 tracrRNA(뉴클레오타이드 4 내지 89)로 프로그래밍하였다. 복합체를 스페이서 2에 상보적인 서열 및 기능적 PAM(4)을 보유하는 이중- 또는 단일-가닥 DNA로 배양하였다. DNA의 상보적 또는 비상보적 가닥은 5'-방사선표지되었고, 비표지된 파트너 가닥으로 어닐링시켰다. nt, 뉴클레오타이드. 도 17, b 및 c 참조. (C) 도 10a로부터 절단 생성물의 서열 분석. 서열분석 반응에서 프라이머 확대 종료는 절단 부위의 위치를 나타낸다. 3' 말단 A 돌출(별표)은 서열분석 반응의 인공물이다. 도 19, a 및 c 참조. (D) 도 10b로부터의 절단 생성물을 표적 DNA 듀플렉스의 상보적 및 비상보적 가닥으로부터 유도된 5' 말단-표지된 크기 마커와 함께 분석하였다. M, 마커; P, 절단 생성물. 도 19, b 및 c 참조. (E) tracrRNA, crRNA-sp2 및 프로토스페이서 2 DNA 서열의 도식적 묘사. tracrRNA에 대한 crRNA 상보성 영역(윗줄) 및 프로토스페이서 DNA(밑줄)가 표현된다. PAM 서열은 표지되고; (C) 및 (D)에서 맵핑된 절단 부위는 화이트 충전된 화살표(C), 블랙 충전된 화살표[(D), 상보적 가닥] 및 블랙 막대[(D), 비상보적 가닥]로 표시된다.
도 15는 유형 II RNA-매개된 CRISPR/Cas 면역 경로를 묘사한다. 발현 및 간섭 단계가 도면에 표시된다. 유형 II CRISPR/Cas 유전자 좌는 단백질을 인코딩하는 4개의 유전자, Cas9, Cas1, Cas2 및 Csn2의 오페론, 독특한 게놈-표적화 스페이서(다이아몬드)가 산재된 동일한 반복체(블랙 직사각형)가 따르는 리더 서열 및 트랜스-활성화 tracrRNA를 인코딩하는 서열로 이루어진 CRISPR 어레이로 구성되어 있다. 에스. 파이오제네스 SF370(수탁 번호 NC_002737)(4)의 유형 II CRISPR/Cas 유전자 좌가 여기에 제시된다. 이 유전자 좌에서 실험적으로 확인된 프로모터 및 전사 종결자가 표시된다(4). CRISPR 어레이는 유형 II 시스템(4)에 특이적인 성숙 과정을 경험하는 전구체 CRISPR RNA(전-crRNA) 분자로서 전사된다. 에스. 파이오제네스 SF370에서, tracrRNA는 전-crRNA의 각 반복체에 대한 상보성을 갖는 길이 171 및 89nt의 2개의 1차 전사체로서 전사된다. 제 1 처리 이벤트는 tracrRNA 대 전-crRNA의 짝짓기, Cas9 단백질의 존재하에 하우스키핑 엔도리보뉴클레아제 RNase III에 의해 인식되고 절단되는 듀플렉스 RNA의 형성을 포함한다. 듀플렉스 RNA의 RNase III-매개된 절단은 75-nt 처리된 tracrRNA 및 반복 서열의 부분에 의해 측면이 접해진 하나의 스페이서의 서열을 함유하는 중심 영역으로 이루어진 66-nt 중간체 crRNA를 생성한다. 공지되지 않은 리보뉴클레아제(들)에 의해 매개된 제 2 처리 이벤트는 5'-말단 스페이서 유도된 가이드 서열 및 반복체 유도된 3'-말단 서열로 이루어진 길이 39 내지 42nt의 성숙한 crRNA의 형성을 유도한다. 제 1 및 제 2 처리 이벤트에 이어, 성숙한 tracrRNA는 성숙한 crRNA과 쌍으로 되고, Cas9 단백질에 결합되어 잔류한다. 이러한 3원 복합체에서, 이중 tracrRNA:crRNA 구조는 동족 표적 DNA에 엔도뉴클레아제 Cas9를 지도하는 가이드 RNA로서 작용한다. Cas9-tracrRNA:crRNA 복합체에 의한 표적 인식은 표적 DNA 중의 프로토스페이서 서열과 crRNA 중의 스페이서 유도된 서열 사이의 상동성에 대해 침입성 DNA 분자를 스캐닝하여 개시한다. DNA 프로토스페이서-crRNA 스페이서 상보성 이외에, DNA 표적화는 프로토스페이서에 인접한 짧은 모티프(NGG, N은 임의의 뉴클레오타이드일 수 있다)(프로토스페이서 인접 모티프 - PAM)의 존재를 필요로 한다. 이중-RNA와 프로토스페이서 서열 사이의 짝짓기에 이어, R-루프가 형성되고, Cas9를 후속적으로 DNA 중의 이중-가닥 파괴(DSB)를 도입한다. Cas9에 의한 표적 DNA의 절단은 단백질 중의 2개의 촉매 도메인을 필요로 한다. PAM에 대해 특이적 부위에서, HNH 도메인은 DNA의 상보적 가닥을 절단하는 반면, RuvC-형 도메인은 비상보적 가닥을 절단한다.
도 16 (A) 에스. 파이오제네스 Cas9는 N-말단 His6-MBP 태그를 함유하는 융합 단백질로서 이. 콜라이에서 발현되고, 친화성, 이온 교환 및 크기 배제 크로마토그래피 단계의 조합으로 정제시켰다. 친화성 태그는 친화도 정제 단계 이후 TEV 프로테아제 절단으로 제거하였다. 슈퍼덱스 200(16/60) 컬럼 상에서 최종 크기 배제 크로마토그래피 단계의 크로마토그램이 제시된다. Cas9는 280 및 260nm에서의 흡광도 비로 판단되는 바와 같이, 오염성 핵산이 없는 단일 단량체성 피크로서 용출시킨다. 삽입; 용출된 분획을 10% 폴리아크릴아미드 겔 상에서 SDS-PAGE로 분해시키고, 심플리블루 안전 염료(Invitrogen)로 염색하였다. (B) 정제된 Cas9 오쏘로그의 SDS-PAGE 분석. Cas9 오쏘로그는 보충 물질 및 방법에서 기술된 바와 같이 정제시켰다. 각 정제된 Cas9 2.5㎍을 4 내지 20% 구배 폴리아크릴아미드 겔 상에서 분석하였고, 심플리블루 안전 염료로 염색하였다.
도 17(또한 도 10 참조). 프로토스페이서 1 서열은 에스. 파이오제네스 SF370 crRNAsp1(4)의 표적인 에스. 파이오제네스 SF370 (M1) SPy_0700으로부터 유래한다. 여기서, 프로토스페이서 1 서열은 PAM을 비기능적 서열(TTG)로부터 기능적 서열(TGG)로 변경하여 조작하였다. 프로토스페이서 4 서열은 에스. 파이오제네스 SF370 crRNA-sp4(4)의 표적인 에스. 파이오제네스 MGAS10750 (M4) MGAS10750_Spy1285로부터 유래한다. (A) 동족 tracrRNA:crRNA 듀플렉스에 의해 유도된 프로토스페이서 1 플라스미드 DNA 절단. 절단 생성물은 아가로스 겔 전기영동에 의해 분해시키고, 브롬화에티듐 염색으로 가시화하였다. M, DNA 마커; 염기쌍에서 분획 크기가 명시된다. (B) 동족 tracrRNA:crRNA-sp1 듀플렉스에 의해 유도된 프로토스페이서 1 올리고뉴클레오타이드 DNA 절단. 절단 생성물은 변성 폴리아크릴아미드 겔 전기영동에 의해 분해시키고, 포스포르이미지화로 가시화하였다. 뉴클레오타이드 중의 단편 크기가 명시된다. (C) 동족 tracrRNA:crRNA-sp4 듀플렉스에 의해 유도된 프로토스페이서 4 올리고뉴클레오타이드 DNA 절단. 절단 생성물은 변성 폴리아크릴아미드 겔 전기영동에 의해 분해시키고, 포스포르이미지화로 가시화하였다. 뉴클레오타이드 중의 단편 크기가 명시된다. (A, B, C) (A)에서 실험은 도 10A에서와 같이 수행하였고, (B) 및 (C)에서의 실험은 도 10B에서와 같이 수행하였다. (B, C) tracrRNA:crRNA 표적 DNA 상호작용의 도식이 이하 제시된다. tracrRNA에 대한 crRNA 상보성 영역 및 프로토스페이서 DNA를 각각 윗줄 및 밑줄친다. PAM 서열은 표지된다.
도 18(또한 도 10 참조). (A) 프로토스페이서 2 플라스미드 DNA는 상이한 농도의 Mg2+, Mn2+, Ca2+, Zn2+, Co2+, Ni2+ 또는 Cu2+의 존재하에 tracrRNA:crRNA-sp2로 복합체된 Cas9로 배양하였다. 절단 생성물은 아가로스 겔 전기영동으로 분해시키고, 브롬화에티듐 염색으로 가시화하였다. 플라스미드 형태가 명시된다. (B) PAM 모티프를 함유하는 프로토스페이서 4 올리고뉴클레오타이드 DNA 듀플렉스를 어닐링시키고, 두 5' 말단에서 방사선표지 전에 겔-정제시켰다. 듀플렉스(10nM 최종 농도)를 tracrRNA(뉴클레오타이드 23-89) 및 crRNAsp4(500nM 최종 농도. 1:1)로 프로그래밍된 Cas9로 배양하였다. 명시된 시점(분)에서, 10㎕ 분취량의 절단 반응물을 0.025% SDS 및 5mM EDTA를 함유하는 포름아미드 완충제로 급냉시키고, 도 10B에서와 같이 변성 폴리아크릴아미드 겔 전기영동으로 분석하였다. 뉴클레오타이드 중의 크기가 명시된다.
도 19 (A) 프로토스페이서 1 플라스미드 DNA 절단의 맵핑. 도 17a로부터의 절단 생성물을 도 10c에서와 같이 서열분석하여 분석하였다. 3' 말단 A 돌출(별표)이 서열분석 반응의 인공물임을 주의한다. (B) 프로토스페이서 4 올리고뉴클레오타이드 DNA 절단의 맵핑. 도 17c로부터의 절단 생성물을 프로토스페이서 4 듀플렉스 DNA의 상보적 및 비상보적 가닥으로부터 유도된 5' 말단표지된 올리고뉴클레오타이드 크기 마커와 함께 변성 폴리아크릴아미드 겔 전기영동에 의해 분석하였다. M, 마커; P, 절단 생성물. 레인 1-2: 상보적 가닥. 레인 3-4: 비상보적 가닥. 뉴클레오타이드 중의 단편 크기가 명시된다. (C) tracrRNA, crRNA-sp1 및 프로토스페이서 1 DNA 서열(상단부) 및 tracrRNA, crRNAsp4 및 프로토스페이서 4 DNA 서열(하단부)의 도식적 묘사. tracrRNA:crRNA는 crRNA-프로토스페이서 DNA 짝짓기를 통해 상보적 프로토스페이서 DNA로 유도된 이중-RNA 구조를 형성한다. tracrRNA에 상보적인 crRNA의 영역 및 프로토스페이서 DNA를 각각 윗줄 및 밑줄친다. (A)(상단부) 및 (B)(하단부)에서 맵핑된 상보적 및 비상보적 DNA 가닥 중의 절단 부위는 각각 그 서열 위 화살표(A 및 B, 상보적 가닥) 및 블랙 막대(B, 비상보적 가닥)로 나타낸다.
도 20 (A) 상이한 RNA 전-어닐링 및 단백질-RNA 전-배양 조건하에 CAs9의 단일 턴오버 동력학. 프로토스페이서 2 플라스미드 DNA는 전-어닐링된 tracrRNA:crRNA-sp2로 전-배양된 Cas9(○), 전-어닐링된 tracrRNA:crRNA-sp2로 전-배양되지 않은 Cas9(●), 전-어닐링되지 않은 tracrRNA 및 crRNA-sp2로 전-배양된 Cas9(□) 또는 전-어닐링되지 않은 RNA로 전-배양되지 않은 Cas9(■)로 배양하였다. 절단 활성은 시간 의존 방식으로 모니터링하고, 아가로스 겔 전기영동에 이어 브롬화에티듐 염색으로 분석하였다. 3개의 독립적 실험으로부터 평균 절단율을 시간(분)에 대해 플롯팅하고, 비선형 회귀로 적합화했다. 계산된 절단 속도(kobs)는 표에 제시한다. 결과는, RNA에 대한 Cas9의 결합이 시험된 조건하에 속도 제한적이지 않음을 제시한다. 플라스미드 형태가 명시된다. 수득된 kobs 값은 전형적으로 1 내지 10/min 정도인 제한 엔도뉴클레오타이드의 값과 비교할만하다(45-47). (B) Cas9는 다중 턴오버 엔도뉴클레아제이다. 듀플렉스화 tracrRNA:crRNA-sp2(1nM, 1:1:1- 그래프에서 회색선으로 명시됨)가 부하된 Cas9는 5배 과량의 천연 프로토스페이서 2 플라스미드 DNA로 배양하였다. 절단은 규정된 시간 간격으로(0 내지 120분) 반응으로부터 샘플을 회수하여 모니터링한 후, 아가로스 겔 전기영동 분석(상단부) 및 절단 생성물 양(nM)의 측정(하단부)이 이어진다. 3개의 독립적 실험의 표준편차가 명시된다. 조사된 시간 간격에서, 1nM Cas9는 약 2.5nM 플라스미드 DNA를 절단할 수 있었다.
각각의 Cas9 뉴클레아제 도메인은 하나의 DNA 가닥을 절단한다
Cas9는 HNH 및 RuvC 엔도뉴클레아제 둘 다에 상동성인 도메인을 함유한다(도 11a 및 도 3)(21-23, 27, 28). 본 발명자들은 HNH 또는 RuvC-형 도메인의 촉매적 잔기에서 불활성화 점 돌연변이를 함유하는 Cas9 변이체를 설계하고 정제시켰다(도 11a 및 도 3)(23,27). 천연 플라스미드 DNA에 의한 이러한 변이체 Cas9 단백질의 배양은 이중-RNA-유도된 돌연변이체 Cas9 단백질은 닉화 개방 원형 플라스미드를 수득한 반면, WT Cas9 단백질-tracrRNA:crRNA 복합체는 선형 DNA 생성물을 생산한다는 것을 보여주었다(도 10a 및 11a 및 도 17a 및 25a). 이 결과는, Cas9 HNH 및 RuvC-형 도메인이 각각 하나의 플라스미드 DNA 가닥을 절단시킨다는 것을 나타낸다. 표적 DNA의 가닥이 각각의 Cas9 촉매 도메인에 의해 절단되는지를 결정하기 위해, 본 발명자들은 상보적 또는 비상보적 가닥이 이의 5' 말단에서 방사선표지된 짧은 dsDNA 기질로 돌연변이체 Cas9-tracrRNA:crRNA 복합체를 배양하였다. 생성되는 절단 생성물은, Cas9 HNH 도메인이 상보적 DNA 가닥을 절단하는 반면, Cas9 RuvC-형 도메인이 비상보적 DNA 가닥을 절단한다는 것을 나타냈다(도 11b 및 도 21b).
도 11 (A) (상단부) 도메인 돌연변이의 위치를 나타내는 Cas9 도메인 구조의 도시적 묘사. D10A, Asp10→Ala10; H840A; His840→Ala840. WT 또는 뉴클레아제 돌연변이체 Cas9 단백질과 tracrRNA:crRNA-sp2의 복합체를 도 10a에서와 같이 엔도뉴클레아제 활성에 대해 검정하였다. (B) WT Cas9 또는 뉴클레아제 도메인 돌연변이체와 tracrRNA 및 crRNA-sp2의 복합체를 도 10b에서와 같이 활성에 대해 시험하였다.
도 3 에스. 파이오제네스의 Cas9의 아미노산 서열(서열번호 8)이 명시된다. 다양한 다른 종으로부터 Cas9/Csn1 단백질은 HNH 및 RuvC 엔도뉴클레아제 둘 다에 상동성인 모티프를 포함하는 2개 도메인을 갖는다. (A) 모티프 1-4(모티프 수는 서열의 좌측에 표기된다)는 에스. 파이오제네스 Cas9/Csn1을 위해 제시되었다. 3개의 예상된 RuvC-형 모티프(1,2,4) 및 예상된 HNH 모티프(3)는 윗줄친다. 이 연구에서 Ala에 의해 치환된 잔기 Asp10 및 His840은 서열 위 별표로 강조된다. 밑줄친 잔기는 상이한 종으로부터 Cas9 단백질 중에서 고도로 보존된다. 밑줄친 잔기 중의 돌연변이는 Cas9 활성에 대한 기능적 결과를 초래할 가능성이 있다. 본 연구에서, 2개의 뉴클레아제형 활성의 결합이 실험적으로 실증된다(도 11 및 도 21)는 것을 주시한다. (B) 모티프 1-4를 포함하는 도메인 1(아미노산 7-166) 및 2(아미노산 731-1003)는 에스. 파이오제네스 Cas9/Csn1에 대해 묘사된다. 추가의 정보를 위해 표 1 및 도 5를 참조한다.
도 21 HNH 또는 RuvC-형 도메인에서 돌연변이를 함유하는 동족 tracrRNA:crRNA-유도된 Cas9 돌연변이체에 의한 프로토스페이서 DNA 절단. (A) 프로토스페이서 1 플라스미드 DNA 절단. 실험은 도 11a에서와 같이 수행하였다. 염기쌍에서 플라스미드 DNA 형태 및 크기가 명시된다. (B) 프로토스페이서 4 올리고뉴클레오타이드 DNA 절단. 실험은 도 11b에서와 같이 수행하였다. 뉴클레오타이드의 크기가 명시된다.
표적 DNA 결합 및 절단을 위한 이중- RNA 요건
tracrRNA는 표적 DNA 결합에 및/또는 표적 인식의 Cas9 다운스트림의 뉴클레아제 활성을 자극하기 위해 필요할 수 있다. 이러한 가능성 사이를 구별하기 위해, 본 발명자들은 crRNA 및/또는 tracrRNA의 존재 또는 부재하에 촉매적 불활성 Cas9에 의한 표적 DNA 결합을 모니터링하기 위해 전기영동 이동도 변화 검정을 사용했다. tracrRNA의 부가는 Cas9에 의한 표적 DNA 결합을 실질적으로 증대시키는 반면, 본 발명자들은 Cas9 단독 또는 Cas9-crRNA와의 작은 특이적 DNA 결합을 관찰하였다(도 22). 이는, tracrRNA가 가능하게는 crRNA를 표적 DNA의 상보적 가닥과의 상호작용에 대해 적절하게 배향함으로써 표적 DNA 인식에 필요하다는 것을 나타낸다. 예상된 tracrRNA:crRNA 2차 구조는 crRNA의 3' 말단에서 22 뉴클레오타이드와 성숙한 tracrRNA의 5' 말단 근처의 세그먼트 사이에 염기 짝짓기를 포함한다(도 10e). 이 상호작용은 상이한 crRNA에서 서열을 변화시키는 crRNA의 5'-말단 20 개의 뉴클레오타이드가 표적 DNA 결합에 이용가능한 구조를 생성한다. crRNA 염기짝짓기 영역의 tracrRNA 다운스트림의 벌크는 추가의 RNA 구조(들)를 형성하고/하거나 Cas9 또는 표적 DNA 부위와 상호작용하는데 자유롭다. tracrRNA의 전체 길이가 부위 특이적 Cas9 촉매된 DNA 절단에 필요한지를 결정하기 위해, 본 발명자들은 전장 성숙 (42-뉴클레오타이드) crRNA 및 이들의 5' 또는 3' 말단에서 tracrRNA 부족 서열의다양한 절단 형태를 사용하여 재구성된 Cas9-tracrRNA:crRNA 복합체를 시험했다. 이러한 복합체들은 짧은 표적 dsDNA를 사용하여 절단에 대해 시험하였다. 천연 서열의 뉴클레오타이드 23 내지 48을 보유한 tracrRNA의 실질적으로 절단된 버젼은 강력한 이중-RNA-유도된 Cas9-촉매된 DNA 절단을 지지할 수 있었다(도 12, a 및 c, 및 도 23, a 및 b). 양쪽 말단으로부터 crRNA의 절단은, tracrRNA의 존재하에 Cas9-촉매된 절단은 3'-말단 10 개의 뉴클레오타이드를 누락한 crRNA로 유발될 수 있었다는 것을 나타냈다(도 12, b 및 c). 대조적으로, crRNA의 5' 말단으로부터 10-뉴클레오타이드 결실가 Cas9에 의한 DNA 절단을 파괴한다(도 12b). 본 발명자들은 또한 에스. 파이오제네스 tracrRNA:crRNA-유도된 DNA 절단을 지지하는 능력에 대해 각종 박테리아 종으로부터 Cas9 오쏘로그를 분석하였다. 밀접하게 관련된 에스. 파이오제네스 Cas9 오쏘로그와 대조적으로, 보다 멀리 관련된 오쏘로그는 절단 반응에서 기능적이지 않았다(도 24). 유사하게, 보다 먼 시스템으로부터 유래하는 tracrRNA:crRNA 듀플렉스에 의해 유도된 에스. 파이오제네스 Cas9는 DNA를 효율적으로 절단할 수 없었다(도 24). DNA의 이중-RNA-유도된 절단의 종 특이성은 Cas9, tracrRNA 및 crRNA 반복체의 공동 진화 뿐만 아니라 특이적 Cas9 오쏘로그와의 삼원 복합체의 형성에 중요한 이중-RNA에서 공지되지 않은 구조 및/또는 서열의 존재를 나타낸다.
박테리아 세포 중의 유형 II CRISPR/Cas 면역성에 대한 프로토스페이서 서열 요건을 조사하기 위해, 본 발명자들은 단일-뉴클레오타이드 돌연변이를 보유하는 일련의 프로토스페이서 함유 플라스미드 DNA를 에스. 파이오제네스에서 형질전환후 그의 유지 및 시험관에서 Cas9에 의해 절단되는 그들의 능력에 대해 분석했다. 프로토스페이서의 5' 말단에 도입된 점 돌연변이와 대조적으로, PAM 및 Cas9 절단 부위에 근접한 영역에서 돌연변이는 생체내에서 허용되지 않았고, 시험관내에서 감소된 플라스미드 절단 효율을 유도했다(도 12d). 본 결과는 생체내 에스. 서모필러스로부터 유형 II CRISPR 시스템에서 선택된 프로토스페이서 회피 돌연변이체의 이전 보고와 일치한다(27,29). 더욱이, 플라스미드 유지 및 절단 결과는 crRNA와의 상호작용 및 Cas9에 의한 후속적인 절단에 중요한 프로토스페이서 서열의 3' 말단에 위치된 "씨드"의 존재를 암시한다. 이 개념을 지원하기 위해, Cas9는 crRNA에 대해 상보적인 DNA 가닥 하이브리드화를 증대시키고; 이 증대는 crRNA 표적화 서열의 3'-말단 영역에서 가장 강했다(도 25a 내지 c). 이 발견을 보강하여, crRNA와 PAM에 인접하는 표적 DNA 부위 사이에 적어도 13 염기쌍의 인접 스트레치가 효율적인 표적 절단에 필요한 반면, 프로토스페이서의 5'-말단 영역에 6개 이하의 인접 불일치가 허용된다(도 12e). 이러한 발견은 아르고노트 단백질(30,31) 및 캐스케이드 및 Csy CRISPR 복합체(13,14)에서 표적 핵산 인식에 대해 이미 관찰된 씨드-서열 요건을 암시한다.
도 12 (A) Cas9-tracrRNA:crRNA 복합체는 42-뉴클레오타이드 crRNA-sp2 및 끝이 절단된 tracrRNA 작제물을 사용하여 재구성하고, 도 10b에서와 같이 절단 활성에 대해 검정하였다. (B) 전장 tracrRNA 및 crRNA-sp2 절단으로 프로그래밍된 Cas9는 (A)에서와 같이 활성에 대해 검정하였다. (C) Cas9-매개된 DNA 절단을 유도할 수 있는 tracrRNA 및 crRNA의 최소 영역(음영 영역). (D) 명시된 점 돌연변이를 갖는 WT 또는 돌연변이체 프로토스페이서 2 서열을 함유하는 플라스미드를 도 10a에서와 같이 프로그래밍된 Cas9에 의해 시험관내에서 절단하고, WT 또는 전-creRNA-결핍된 에스. 파이오제네스의 형질전환 검정에 사용하였다. 형질전환 효율은 플라스미드 DNA 1㎍당 콜로니-형성 단위(CFU)로서 계산되었다. 오차 막대는 3개의 생물학적 복제물에 대한 SD를 나타낸다. (E) 변화하는 정도의 crRNA-표적 DNA 불일치(하단부)와 함께 WT 및 돌연변이체 프로토스페이서 2 삽입체를 함유하는 플라스미드는 프로그래밍된 Cas9(상단부)에 의해 시험관내에서 절단되었다. 절단 반응물은 XmnI로 추가로 소화되었다. 1880- 및 800-bp 단편은 Cas9-생성된 절단 생성물이다. M, DNA 마커.
도 22 전기영동 이동도 변화 검정은 프로토스페이서 4 표적 DNA 듀플렉스 및 Cas9(뉴클레아제 도메인 불활성화 돌연변이 D10A 및 H840 함유)를 단독으로또는 crRNA-sp4,tracrRNA(75nt)의 존재하에 또는 이들 둘 다하에 사용하여 수행하였다. 표적 DNA 듀플렉스는 두 5' 말단에서 방사선표지하였다. Cas9(D10/H840A) 및 복합체는 1nM 내지 1μM로 적정하였다. 결합은 8% 천연 폴리아크릴아미드 겔 전기영동으로 분석하고, 포스포르이미지화로 가시화하였다. Cas9 단독이 보통의 친화성으로 표적 DNA와 결합한다는 것을 주시한다. 이 결합은 crRNA의 첨가로 영향을 받지 않고, 이는 dsDNA와 서열 비특이적 상호작용을 나타낸다는 것을 시사한다. 더욱이, 이 상호작용은 crRNA의 부재하에 tracrRNA 단독으로 더 성공할 수 있다. crRNA 및 tracrRNA 둘 다의 존재하에, 표적 DNA 결합은 실질적으로 증대되고, 특이적 표적 DNA 인식을 나타내는 뚜렷한 전기영동 이동도를 갖는 종을 산출한다.
도 23 crRNA 쌍으로 된 영역의 일부 및 다운스트림 영역의 일부를 포함하는 tracrRNA의 단편은 Cas9에 의한 프로토스페이서 올리고뉴클레오타이드 DNA의 직접 절단에 충분하다. (A) 성숙한 동족 CrRNA 및 다양한 tracrRNA 단편으로 유도된 Cas9에 의한 프로토스페이서 1 올리고뉴클레오타이드 DNA 절단 및 (B) 프로토스페이서 4 올리고뉴클레오타이드 DNA 절단. (A,B) 뉴클레오타이드의 크기가 명시된다.
도 24 에스. 파이 오제네스로부터 Cas9와 같이, 그람 양성 박테리아 엘. 이노쿠아 및 에스. 서모필러스로부터 밀접하게 관련된 Cas9 오쏘로그는 에스. 파이오제네스로부터 tracrRNA:crRNA로 표적화될 경우, 프로토스페이서 DNA를 절단한다. 그러나, 동일한 조건 하에서, 그람 음성 박테이라 씨. 제주니 및 엔. 메닌자이티디스로부터의 덜 밀접하게 관련된 Cas9 오쏘로그의 의한 DNA 절단은 관찰되지 않는다. Spy, 에스. 파이오제네스 SF370 (수탁 번호 NC_002737); Sth, 에스. 서모필러스 LMD-9 (STER_1477 Cas9 오쏘로그; 수탁 번호 NC_008532); Lin, 엘. 이노쿠아 Clip11262 (수탁 번호 NC_003212); Cje, 씨. 제주니 NCTC 11168 (수탁 번호 NC_002163); Nme, 엔. 메닌자이티디스 A Z2491 (수탁 번호 NC_003116). (A) 프로토스페이서 플라스미드 DNA의 절단. 프로토스페이서 2 플라스미드 DNA(300ng)를 상이한 종으로부터 하이브리드 tracrRNA:crRNA-sp2 듀플렉스(500nM, 1:1)로 유도된 상이한 Cas9 오쏘로그(500nM)에 의한 절단에 적용했다. RNA 듀플렉스를 설계하기 위해, 본 발명자들은 이전에 공개된 노던 블랏 데이터를 기반으로 하는 엘. 이노쿠아 및 엔. 메닌자이티디스로부터의 tracrRNA 서열을 예상한다(4). 이중-하이브리드 RNA 듀플렉스는 종 특이적 tracrRNA 및 이종기원 crRNA로 이루어진다. 이종기원 crRNA 서열은 3' 말단에서 엘. 이노쿠아 또는 엔. 메닌자이티디스 tracrRNA-결합 반복 서열에 융합된 5' 말단에 에스. 파이오제네스 DNA-표적화 sp2 서열을 함유하도록 조작되었다. 엔. 메닌자이티디스 또는 씨. 제주니로부터가 아니라 에스. 서모필러스 및 엘. 이노쿠아로부터의 Cas9 오쏘로그는 에스. 파이오제네스 tracrRNA:crRNA-sp2에 의해 유도되어 약간 감소된 효율에도 불구하고 프로토스페이서 2 플라스미드 DNA를 절단할 수 있다. 유사하게, 하이브리드 엘. 이노쿠아 tracrRNA:crRNA-sp2는 에스. 파이오제네스 Cas9를 유도하여 고효율로 표적 DNA를 절단할 수 있는 반면, 하이브리드 엔. 메닌자이티디스 tracrRNA:crRNA-sp2는 에스. 파이오제네스 Cas9에 의한 단지 약간의 DNA 절단 활성을 유발한다. 대조군으로서, 엔. 메닌자이티디스 및 엘. 이노쿠아 Cas9 오쏘로그는 동족 하이브리드 tracrRNA:crRNA-sp2에 의해 유도될 경우, 프로토스페이서 2 플라스미드 DNA를 절단한다. 상기에서 언급된 바와 같이, 엔. 메닌자이티디스의 tracrRNA 서열이 단지 예상되고, RNA 서열분석에 의해 아직 확인되지 않았음을 주시한다. 따라서, 절단 저효율은 Cas9 오쏘로그의 저활성 또는 비최적으로 설계된 tracrRNA 서열의 사용 결과일 수 있었다. (B) 프로토스페이서 올리고뉴클레오타이드 DNA의 절단. 비표지된 비상보적 가닥 올리고뉴클레오타이드(프로토스페이서 1)(10nM)(좌측)로 전-어닐링된 5'-말단 방사선활동적으로 표지된 상보적 가닥 올리고뉴클레오타이드(10nM) 또는 비표지된 상보적 가닥 올리고뉴클레오타이드(10nM)(우측)(프로토스페이서 1)로 전-어닐링된 5'-말단 방사선활동적으로 표지된 비상보적 가닥 올리고뉴클레오타이드(10nM)를 에스. 파이오제네스(500nM, 1:1)로부터의 tracrRNA:crRNA-sp1 듀플렉스에 의해 유도된 다양한 Cas9 오쏘로그(500nM)에 의한 절단에 적용하였다. 엔. 메닌자이티디스 또는 씨. 제주니로부터가 아니라 에스. 서모필러스 및 엘. 이노쿠아로부터의 Cas9 오쏘로그는 에스. 파이오제네스 동족 이중-RNA에 의해 유도되어 감소된 효율에도 불구하고 프로토스페이서 올리고뉴클레오타이드 DNA를 절단할 수 있다. 상보적 DNA 가닥 상의 절단 부위는 모두 3개 오쏘로그에 대해 동일하다는 것을 주시한다. 비상보적 가닥의 절단은 명확한 위치에서 발생한다. (C) Cas9 오쏘로그의 아미노산 서열 동일성. 에스. 파이오제네스, 에스. 서모필러스 및 엘. 이노쿠아 Cas9 오쏘로그는 높은 비율의 아미노산 동일성을 공유한다. 대조적으로, 씨. 제주니 및 엔. 메닌자이티디스 Cas9 단백질은 서열 및 길이가 상이하다(약 300 내지 400 아미노산 더 짧음). (D) 에스. 파이 오제네스(실험적으로 확인됨, (4)), 엘. 이노쿠아(예상됨) 또는 엔. 메닌자이티디스(예상됨)로부터 상응하는 tracrRNA 오쏘로그를 갖는 조작된 종-특이적 이종기원 crRNA 서열의 공동-폴딩. tracrRNAs;crRNA 스페이서 2 단편; 및 crRNA 반복체 단편을 추적하고, 표지화한다. 엘. 이노쿠아 및 에스. 파이오제네스 하이브리드 tracrRNA:crRNA-sp2 듀플렉스는 엔. 메닌자이티디스 하이브리드 tracrRNA:crRNA와 상이함에도 불구하고, 매우 유사한 구조적 특성을 공유한다. (A) 및 (B)에서 상기한 절단 데이터와 함께, 공동-폴딩 예측은 Cas9-tracrRNA:crRNA에 의한 표적 DNA의 종 특이성 절단이 동족 Cas9 오쏘로그에 의해 특이적으로 인식된 tracrRNA:crRNA 듀플렉스에서 여전히 공지되지 않은 구조적 특징에 의해 지시된다는 것을 나타낼 것이다. (A) 및 (B)에서 관찰된 절단의 종-특이성은 Cas9 대 이중-tracrRNA:crRNA의 결합 수준에서 발생한다고 예상되었다. 표적 DNA의 이중-RNA 유도된 Cas9 절단은 종 특이적일 수 있다. Cas9 단백질 및 tracrRNA:crRNA 듀플렉스 중에서 다양성/진화 정도에 따라, Cas9 및 이중-RNA 오쏘로그는 부분적으로 교환가능하다.
도 25 DNA-표적화 영역 및 tracrRNA-결합 영역을 포함하는 crRNA 중의 영역에 상보적인 일련의 8-뉴클레오타이드 DNA 프로브를 tracrRNA:crRNA 듀플렉스 및 Cas9-tracrRNA:crRNA 3원 복합체의 문맥에 있어서, crRNA로 하이브리드화하는 그들의 능력에 대해 분석하였다. (A) 검정에 사용된 DNA 프로브의 서열 및 crRNA-sp4 중의 그들의 결합 부위의 도식적 묘사. (B-C) tracrRNA:crRNA-sp4 또는 Cas9-tracrRNA:crRNA-sp4를 갖는 표적 DNA 프로브의 전기영동 이동도 변화 검정. tracrRNA(15-89) 작제물이 실험에 사용되었다. 표적 올리고뉴클레오타이드 DNA에 대한 듀플렉스 또는 복합체의 결합을 16% 천연 폴리아크릴아미드 겔 상에서 분석하고, 포스포르이미지화로 가시화하였다.
짧은 서열 모티프는 R-루프 형성을 지시한다
다중 CRISPR/Cas 시스템에서, 자기 대 비자기 인식은 PAM(27, 29, 32-34)으로서 칭명되는, 외래 게놈에 보존된 짧은 서열 모티프를 포함하는 것으로 나타났다. PAM모티프는 CRISPR/Cas 시스템 유형(32)에 따라 변하는 길이, 그들의 정확한 서열 및 위치의 유일한 소수 염기쌍이다. 에스. 파이오제네스 유형 II 시스템에서, PAM은 표적 DNA(4) 내에서 crRNA 결합 서열의 하나의 염기상 다운스트림을 발생하는 두 G:C 염기쌍을 함유하는 NGG 공통 서열에 일치한다. 형질전환 검정은 GG 모티프가 박테리아 세포(도 26a) 중의 CRISPR/Cas에 의한 프로토스페이서 플라스미드 DNA 제거에 필수적이고, 에스. 서모필러스(27)에서 이전 관찰과 일치한다는 것을 입증하였다. 모티프는 또한 tracrRNA:crRNA-유도된 Cas9(도 26b)에 의한 시험관내 프로토스페이서 플라스미드 절단에 필수적이다. Cas9-tracrRNA:crRNA 복합체에 의한 표적 DNA 절단에서 PAM의 역할을 결정하기 위해, 본 발명자들은 상보적 또는 비상보적 가닥, 또는 둘 다(도 13a)에 대한 PAM 서열에 돌연변이를 함유하는 일련의 dsDNA 듀플렉스를 시험했다. 이러한 기질을 사용하는 절단 검정은 Cas9-촉매된 DNA 절단이 유형 I CRISPR/Cas 시스템(18, 34)에 의한 상보적 가닥 PAM 인식과 대조적으로, DNA의 비상보적 가닥 상의 PAM 서열 중의 돌연변이에 특히 민감하였다는 것을 나타냈다. 표적 단일-가닥 DNA의 절단은 PAM 모티프의 돌연변이에 의해 영향받지 않았다. 이 관찰은 PAM 모티프가 표적 dsDNA의 문맥에서 유일하게 필요하고, 따라서 듀플렉스 풀기, 가닥 침입 및 R-루프 구조의 형성을 허용하는데 필요할 수 있다는 것을 시사한다. 본 발명자들이 프로토스페이서 2 표적 DNA에 존재하지 않는 표준적 PAM의 존재에 기인하여 선택된 상이한 crRNA-표적 DNA 쌍(crRNA-sp4 및 프로토스페이서 4 DNA)을 사용할 경우, 본 발명자들은 PAM의 두 G 뉴클레오타이드가 효율적인 Cas9-촉매된 DNA 절단(도 13b 및 도 26c)에 필요했다는 것을 밝혀냈다. PAM이 정확한 표적 DNA 부위에 Cas9-tracrRNA:crRNA 복합체를 모집하는데 직접적인 역할을 하는지를 결정하기 위해, 본 발명자들은 천연 겔 이동도 변화 검정(도 13c)에 의해 표적 DNA 서열에 대한 복합체의 결합 친화성을 분석하였다. PAM 서열 중의 하나의 G의 돌연변이는 실질적으로 표적 DNA에 대한 Cas9-tracrRNA: crRNA의 친화성을 감소시킨다. 이 발견은 Cas9에 의한 표적 DNA 결합에서 PAM 서열의 역할을 설명한다.
도 13 (A) 이중 RNA-프로그래밍된 Cas9를 도 10b에서와 같이 활성에 대해 시험하였다. 표적 DNA 중의 WT 및 돌연변이체 PAM 서열은 직선으로 명시된다. (B) WT 및 돌연변이체 PAM 모티프를 함유하는 프로토스페이서 4 표적 DNA 듀플렉스(두 5' 말단에서 표지됨)는 tracrRNA:crRNA-sp4(뉴클레오타이드 23 내지 89)로 프로그래밍된 Cas9로 배양하였다. 명시된 시점(분)에서, 분취량의 절단 반응물을 취하여 도 10b에서와 같이 분석하였다. (C) 전기영동 이동도 변화 검정을 RNA-프로그래밍된 Cas9 (D10A/H840A) 및 WT 및 돌연변이된 PAM 모티프를 함유하는 프로토스페이서 4 표적 DNA 듀플렉스[(B)에서와 동일]를 사용하여 수행하였다. Cas9 (D10A/H840A)-RNA 복합체는 100pM 내지 1mM로 적정하였다.
도 26 (A) 프로토스페이서 2 플라스미드 DNA 중의 PAM 서열의 돌연변이는 박테리아 세포에서 유형 II CRISPR/Cas 시스템에 의한 플라스미드 유지의 간섭을 파괴한다. 기능성 또는 돌연변이된 PAM을 갖는 야생형 프로토스페이서 2 플라스미드는 도 12d에서와 같이 야생형(균주 SF370, 또한 EC904로 명명됨) 및 전-crRNA-결핍된 돌연변이체 (EC1479) 에스. 파이오제네스로 형질전환시켰다. PAM 돌연변이는 생체내 유형 II CRISPR/Cas 시스템에 의해 허용되지 않는다. 3개의 생물학적 복제물의 평균 값 및 표준 편차가 제시된다. (B) 프로토스페이서 플라스미드 DNA 중의 PAM 서열의 돌연변이는 Cas9-tracrRNA:crRNA에 의한 절단을 파괴한다. 기능성 또는 돌연변이된 PAM을 갖는 야생형 프로토스페이서 2 플라스미드는 도 10a에서와 같이 Cas9 절단에 적용하였다. PAM 돌연변이체 플라스미드는 Cas9-tracrRNA:crRNA 복합체에 의해 절단되지 않는다. (C) 표준적 PAM 서열의 돌연변이는 박테리아 세포에서 유형 II CRISPR/Cas 시스템에 의한 플라스미드 유지의 간섭을 파괴한다. 기능성 또는 돌연변이된 PAM을 갖는 야생형 프로토스페이서 4 플라스미드는 tracrRNA 및 crRNA-sp2로 프로그래밍된 Cas9로 절단되었다. 절단 반응은 XmnI 제한 엔도뉴클레아제의 존재하에 수행하여 Cas9 절단 생성물을 2개의 단편(약 1880 및 약 800bp)으로서 가시화하였다. 염기쌍에서 단편 크기가 명시된다.
Cas9 는 단일 키메라 RNA 로 프로그래밍될 수 있다
아마 tracrRNA:crRNA 듀플렉스(도 10e 및 12c)의 2차 구조의 시험은 부위 특이적인 Cas9-촉매된 DNA 절단에 필요한 특징이 단일 키메라 RNA에서 포획될 수 있다는 가능성을 제안한다. tracrRNA:crRNA 표적-선택 기전이 사실상 효율적으로 작동하지만, 단일 RNA-유도된 Cas9가 프로그래밍된 DNA 절단 및 게놈 편집(도 1a-b)에 대한 이의 잠재적인 유용성에 기인하여 호의적이다. 본 발명자들은 tracrRNA 및 crRNA 사이에 발생하는 염기-짝짓기 상호작용을 유지하는 헤어핀 구조가 후속되는 5' 말단에 표적 인식 서열을 함유하는 키메라 RNA의 두 버젼을 설계한다(도 14a). 이 단일 전사체는 효과적으로 crRNA의 3' 말단을 tracrRNA의 5' 말단에 융합시키고, 그렇게 함으로써 Cas9에 의한 부위 특이적 DNA 절단을 유도하는데 필요한 이중-RNA 구조를 모방한다. 플라스미드 DNA를 사용하는 절단 검정에서, 본 발명자들은 보다 긴 키메라 RNA가 끝이 잘린 tracrRNA:crRNA 듀플렉스에 대해 관찰된 바와 유사한 방식으로 Cas9-촉매된 DNA 절단을 유도할 수 있다는 것을 관찰하였다(도 14a 및 도 27, a 및 c). 보다 짧은 키메라 RNA는 이 검정에서 효율적으로 작용하지 않고, tracrRNA:crRNA 염기-짝짓기 상호작용의 범위를 넘어 5 내지 12 위치에 존재하는 뉴클레오타이드가 효율적인 Cas9 결합 및/또는 표적 인식에 중요하다는 것을 확인한다. 본 발명자들은 기질로서 짧은 dsDNA를 사용하는 절단 검정에서 유사한 결과를 수득하여 추가로 표적 DNA 중의 절단 부위의 위치가 가이드로서 이중 tracrRNA:crRNA를 사용하여 관찰된 것과 동일하다는 것을 나타낸다(도 14b 및 도 27, b 및 c). 최종적으로, 키메라 RNA의 설계가 보편적으로 이용가능한지를 입증하기 위해, 본 발명자들은 그린-형광 단백질(GFP)(도 28, a 내지 c)을 인코딩하는 유전자 부분을 표적화하기 위해 5개의 상이한 키메라 가이드 RNA를 조작하였고, 시험관내에서 GFP 코딩 서열을 수반하는 플라스미드에 대한 그들의 효능을 시험하였다. 5개 경우 모두에서, 이러한 키메라 RNA로 프로그래밍된 Cas9는 효율적으로 정확한 표적 부위에서 플라스미드를 절단하고(도 14c 및 도 28d), 키메라 RNA의 합리적인 설계가 강력하고, 원칙적으로, 목적하는 임의의 DNA 서열의 표적화를 표적화된 서열에 인접한 GG 디뉴클레오타이드의 존재를 넘어 약간의 구속으로 가능하게 할 수 있음을 나타내었다.
도 1 DNA-표적화 RNA는 이중 가닥 RNA의 스트레치를 포함하는 단일 가닥 "DNA-표적화 세그먼트" 및 "단백질 결합 세그먼트"을 포함한다. (A) DNA-표적화 RNA는 두 개의 별도의 RNA 분자("이중-분자" 또는 "두-분자" DNA-표적화 RNA로서 칭명됨)를 포함할 수 있다. 이중-분자 DNA-표적화 RNA는 "타겟터-RNA" 및 "활성제-RNA"를 포함한다. (B) DNA-표적화 RNA는 단일 RNA 분자("단일-분자" DNA-표적화 RNA로서 칭명됨)를 포함할 수 있다. 단일-분자 DNA-표적화 RNA는 "링커 뉴클레오타이드"를 포함한다.
도 14 (A) 프로토스페이서 4 표적 서열 및 WT PAM을 보유하는 플라스미드를 tracrRNA(4-89):crRNA-sp4 듀플렉스 또는 crRNA의 3' 말단을 GAAA 테트라루프를 갖는 tracrRNA의 5' 말단과 결합시킴으로써 작제된 시험관내-전사된 키메라 RNA로 프로그래밍된 Cas9에 의한 절단에 적용하였다. 절단 반응을 XmnI를 사용하는 제한 맵핑으로 분석하였다. 키메라 RNA A 및 B의 서열은 DNA-표적화(밑줄), crRNA 반복체-유도된 서열(윗줄), 및 tracrRNA-유도된(점선 밑줄) 서열과 함께 제시된다. (B) 프로토스페이서 4 DNA 듀플렉스 절단 반응을 도 10b에서와 같이 수행하였다. (C) GFP 유전자를 표적화하도록 설계된 5개의 키메라 RNA를 사용하여 Cas9를 GFP 유전자 함유 플라스미드를 절단하도록 프로그래밍하였다. 플라스미드 DNA가 Cas9 절단 후 AvrII로 제한 맵핑된 것 이외에는, 플라스미드 절단 반응을 도 12e에서와 같이 수행하였다.
도 27 (A) 단일 키메라 RNA는 동족 프로토스페이서 플라스미드 DNA(프로토스페이서 1 및 프로토스페이서 2)의 Cas9-촉매된 절단을 유도한다. 절단 반응을 XmnI 제한 엔도뉴클레아제의 존재하에 수행하여 Cas9 절단 생성물을 두 개의 단편(약 1880 및 약 800bp)으로서 가시화하였다. 염기쌍에서 단편 크기가 명시된다. (B) 단일 키메라 RNA는 동족 프로토스페이서 올리고뉴클레오타이드 DNA(프로토스페이서 1 및 프로토스페이서 2)의 Cas9-촉매된 절단을 유도한다. 뉴클레오타이드의 단편 크기가 명시된다. (C) 실험에 사용된 키메라 RNA의 도식적 묘사. 키메라 RNA A 및 B의 서열은 crRNA의 5' 프로토스페이서 DNA-표적화 서열(밑줄), crRNA의 tracrRNA-결합 서열(윗줄) 및 tracrRNA-유도된 서열(점선 밑줄)과 함께 제시된다.
도 28 (A) GFP 발현 플라스미드 pCFJ127의 도식적 묘사. GFP 개방 판독 프레임의 표적화 부분은 블랙 화살표로 명시된다. (B) 표적화된 영역의 서열의 클로즈-업. 키메라 RNA로 표적화된 서열은 회색 막대로 제시된다. PAM 디뉴클레오타이드는 박스화된다. 독특한 SalI 제한 부위는 표적 유전자 좌의 60bp 업스트림에 존재한다. (C) 좌측: 표적 DNA 서열은 그들의 인접하는 PAM 모티프와 함께 제시된다. 우측: 키메라 가이드 RNA의 서열. (D) pCFJ127은 명시된 바와 같이 키메라 RNAs GFP1-5로 프로그래밍된 Cas9에 의해 절단되었다. 플라스미드는 SalI로 추가로 소화되고, 반응은 3% 아가로스 겔 상에서 전기영동으로 분석하고 SYBR Safe로 염색하여 가시화하였다.
결론
Cas9 엔도뉴클레아제를 표적 DNA에 부위 특이적 이중 가닥 파괴를 도입하도록 지시하는 이중-RNA 구조를 포함하는 DNA 간섭 기전이 확인되었다. tracrRNA:crRNA-유도된 Cas9 단백질은 표적 DNA 중의 두 가닥을 절단하기 위해 뚜렷한 엔도뉴클레아제 도메인(HNH 및 RuvC-형 도메인)을 사용ㅘ세 한다. Cas9에 의한 표적 인식은 crRNA 중의 씨드 서열 및 DNA 표적 중의 crRNA 결합 영역에 인접한 GG 디뉴클레오타이드-함유 PAM 서열 둘 다를 필요로 한다. 본 발명자들은 추가로 Cas9 엔도뉴클레아제가 목적하는 임의의 dsDNA 서열을 표적화하고 절단하기 위한 단일 전사체로서 조작된 가이드 RNA로 프로그래밍될 수 있다는 것을 보여준다. 상기 시스템은 효율적이고, 다용도성이고, 가이드 키메라 RNA에서 DNA 표적 결합 서열을 변경함으로써 프로그래밍가능하다. 아연-핑거 뉴클레아제 및 전사-활성제-형 효과기 뉴클레아제는 게놈(35-38)을 조작하도록 조작된 인공 효소로서 상당한 관심을 모으고 있다. 이는 유전자 표적화 및 게놈-편집 적용을 촉진시키는 RNA-프로그래밍된 Cas9를 기반으로 하는 대안적인 방법론을 나타낸다.
인용된 참조 문헌
실시예 2: 인간 세포에서 RNA-프로그래밍된 게놈 편집
이하 제공된 데이터는 Cas9가 인간 세포의 핵에서 발현되어 국재화될 수 있고, 그것이 인간 세포에서 단일-가이드 RNA("sgRNA"; Cas9 결합 및 DNA 표적 부위 인식 둘 다에 필요한 특징을 포함함)와 조립된다는 것을 입증한다. 이러한 복합체는 이중 가닥 파괴를 생성할 수 있고, sgRNA 서열에 상보적인 부위에서 Cas9 및 sgRNA 둘 다를 필요로 하는 활성인 게놈 DNA 중의 비-상동성 말단 결합(NHEJ) 회복을 자극할 수 있다. 3' 말단에서 RNA 서열의 확대는 살아 있는 세포에서 DNA 표적화 활성을 증강시킨다. 또한, 형질감염된 세포로부터의 추출물을 사용하는 실험은, Cas9 속에서 sgRNA 어셈블리가 Cas9-매개된 DNA 절단의 제한 인자임을 보여준다. 이들 결과는 RNA-프로그래밍된 게놈 편집이 살아 있는 세포 및 생체내에서 작동한다는 것을 입증한다.
물질 및 방법
플라스미드 설계 및 구성
HA 에피토프에 융합된 스트렙토코쿠스 파이오제네스 Cas9 (잔기 1-1368)를 인코딩하는 서열(아미노산 서열 DAYPYDVPDYASL (서열번호 274)), 핵 국재화 신호 (아미노산 서열 PKKKRKVEDPKKKRKVD (서열번호 275))를 인간 발현을 위해 코돈 최적화하고, GeneArt로 합성하였다. DNA 서열은 서열번호 276이고, 단백질 서열은 서열번호 277이다. 결찰-독립적인 클로닝(LIC)을 사용하여 이 서열을 pcDNA3.1-유도된 GFP 및 mCherry LIC 벡터(각각, 벡터 6D 및 6B, UC Berkeley MacroLab으로부터 수득됨)에 삽입하여 CMV 프로모터의 조절하에 발현된 Cas9-HA-NLS-GFP 및 Cas9-HA-NLS-mCherry 융합을 초래한다. 가이드 sgRNA는 발현 벡터 pSilencer 2.1-U6 puro (Life Technologies) 및 pSuper (Oligoengine)를 사용하여 발현시켰다. RNA 발현 작제물은 RNA-코딩 DNA 서열을 형성하기 위한 상보적 올리고뉴클레오타이드를 어닐링시키고, 어닐링된 DNA 단편을 pSilencer 2.1-U6 puro 중의 BamHI 및 HindIII 부위와 pSuper 중의 BgiII 및 HindIII 부위 사이에서 결찰시켜 생성시켰다.
세포 배양 조건 및 DNA 형질감염
HEK293T 세포를 5% CO2를 포함하는 37℃ 가습된 배양기에서 10% 소 태아 혈청(FBS)이 보충된 둘베코 변형 이글 배지(DMEM)에서 유지시켰다. 세포를 추천된 프로토콜을 갖는 X-tremeGENE DNA 형질감염 시약(Roche) 또는 Turbofect 형질감염 시약(Thermo Scientific)을 사용하여 플라스미드 DNA로 일시적으로 형질감염시켰다. 간단히 말해서, HEK293T 세포를 6-웰 플레이트에서 0.5㎍의 Cas9 발현 플라스미드 및 2.0㎍의 RNA 발현 플라스미드를 사용하여 60-80% 밀집도로 형질감염시켰다. 형질감염 효율성은 형광 현미경검사에 의해 관찰된 GFP-양성 세포의 분획을 기반으로 하여, Tubofect(도 29e 및 37a-b)의 경우 30 내지 50% 및 X-tremegene(도 31b)의 경우 80 내지 90%인 것으로 추정되었다. 형질감염 후 48시간에, 세포를 포스페이트 완충된 염수(PBS)로 세척하고, 250㎕의 세포용해 완충제(20 mM Hepes pH 7.5, 100mM 염화칼륨(KCl), 5mM 염화마그네슘(MgCl2), 1mM 디티오트레이톨(DTT), 5% 글리세롤, 0.1% 트리톤 X-100, 로슈(Roche) 프로테아제 억제제 칵테일 보충됨)를 적용하여 용해시킨 다음, 10분 동안 4℃에서 진동시켰다. 수득한 세포 용해물을 추가 분석을 위해 분취량으로 분할했다. 게놈 DNA는 제조업자의 프로토콜에 따라 DNeasy 혈액 및 조직 키트(Qiagen)를 사용하여 200㎕ 세포 용해물로부터 단리시켰다.
Cas9 발현의 웨스턴 블롯 분석
Cas9-HA-NLS-GFP 발현 플라스미드로 형질감염된 HEK293T를 수거하고, 상기한 바와 같이 형질감염 후 48시간에 용해시켰다. 5㎕의 용해물을 PVDF 막 상에 압지된 10% SDS 폴리아크릴아미드 겔 상에서 전기영동시키고, HRP-접합된 항-HA 항체(시그마, 1x PBS 중의 1:1000 희석)로 탐색하였다.
감독자 검정
감독자 검정은 이전에 기재된 바와 같이 수행하였다[10,12,13]. 간단히 말해서, 인간 클라트린 경쇄 A(CLTA) 유전자 좌는 고충실도 폴리머라제, 헤르쿨라제 II 융합 DNA 폴리머라제(Agilent Technologies) 및 전방 프라이머 5'-GCAGCAGAAGAAGCCTTTGT-3'(서열번호 //) 및 역방 프라이머 5'-TTCCTCCTCTCCCTCCTCTC-3'(서열번호 //)를 사용하여 200ng의 게놈 DNA로부터 PCR 증폭시켰다. 이어서, 300ng의 360bp 앰플리콘을 95℃로 가열하여 변성시키고, 열 블록을 사용하여 서서히 재어닐링시켜 야생형 및 돌연변이체 DNA 가닥을 랜덤하게 재하이브리드화하였다. 이어서, 샘플을 42℃에서 1시간 동안 Cel-1 뉴클레아제(감독자 키트, 트랜스게놈)로 배양하였다. Cel-1은 불일치(야생형:돌연변이체 하이브리드화)를 함유하는 DNA 나선을 인식하고 절단한다. Cel-1 뉴클레아제 소화 생성물을 10% 아크릴아미드 겔 상에서 분리시키고, SYBR Safe(Life Technologies)로 염색하여 가시화하였다. 절단 밴드의 정량화를 이미지랩 소프트웨어(Bio-Rad)를 사용하여 수행하였다. 절단율은 절단 생성물의 평균 강도((160 내지 200bps)를 절단되지 않은 PCR 생성물(360bp) 및 절단 생성물의 강도의 합으로 나누어 결정하였다.
시험관내 전사
가이드 RNA는 재조합 T7 RNA 폴리머라제를 사용하여 시험관내 전사하였고, DNA 템플레이트는 상보적 합성 올리고뉴클레오타이드를 이전에 기재된 바와 같이 어닐링시켜 생성하였다[14]. RNA는 7M 우레아 변성 아크릴아미드 겔 상에서 전기영동으로 정제시키고, 에탄올 침전시키고, DEPC-처리된 물에 용해시켰다.
노던 블랏 분석
RNA를 미르바나 소-RNA 단리 키트(Ambion)를 사용하여 HEK293T 세포로부터 정제시켰다. 각 샘플을 위해, 800ng의 RNA를 RNA 부하 완충제(0.5X TBE (pH7.5), 0.5mg/ml 브로모페놀 블루, 0.5mg 크실렌 시아놀 및 47% 포름아미드) 중에서 70℃에서 10분 동안 변성 후 10% 우레아-PAGE 겔 상에서 분리시켰다. 브로모페놀 블루 염료가 겔의 바닥에 도달할 때까지 0.5X TBE 완충제 중에서 10W에서 전기영동 후, 샘플을 0.5X TBE 중에서 1.5시간 동안 20V에서 니트란(Nytran) 막 상에서 전자블롯화하였다. 이동된 RNA를 니트란 막 상에서 UV-가교결합제(Strategene) 중의 니트란 막 상에서 가교결합시키고, 45℃에서 3시간 동안 40% 포름아미드, 5X SSC, 3X 던하르트(Dernhardt's)(0.1% 각각의 피콜, 폴리비닐피롤리돈 및 BSA) 및 200㎍/ml 연어 정자 DNA를 함유하는 완충제에서 전-하이브리드화하였다. 전-하이브리드화된 막을 1백만cpm/ml에서 5'-32P-표지된 안티센스 DNA 올리고 프로브가 보충된 전하이브리드화 완충제에서 밤새 배양하였다. SSC 완충제(0.2X SCC로 최종 세척)로 수회 세척 후, 막을 포스포르이미지화로 이미지화하였다.
시험관내 절단 검정
세포 용해물을 상기한 바와 같이 제조하고, CLTA-RFP 공여체 플라스미드[10]로 배양하였다. 절단 반응은 총 용적 20㎕로 수행하였고, 10㎕ 용해물, 2㎕의 5x 절단 완충제(100 mM HEPES pH 7.5, 500 mM KCl, 25 mM MgCl2, 5 mM DTT, 25% 글리세롤) 및 300ng 플라스미드를 함유하였다. 명시될 경우, 반응물은 10pmol의 시험관내 전사된 CLTA1 sgRNA로 보충되었다. 반응물을 37℃에서 1시간 동안 배양하고, 후속적으로 37℃에서 추가로 30분 동안 10U의 XhoI(NEB)로 소화시켰다. 반응은 프로테이나제 K(Thermo Scientific)를 첨가하여 정지시키고, 37℃에서 15분 동안 배양하였다. 절단 생성물을 1% 아가로스 겔 상에서 전기영동으로 분석하였고, SYBR Safe로 염색하였다. 약 2230 및 약 3100bp 단편의 존재는 Cas9-매개된 절단을 나타낸다.
결과
Cas9가 살아 있는 세포에서 게놈 DNA를 절단하도록 프로그래밍될 수 있는지를 시험하기 위해, Cas9는 인간 클라트린 경쇄(CLTA) 유전자를 표적화하기 위해 소화된 sgRNA와 함께 공동-발현시켰다. CLTA 게놈 유전자 좌는 이전에 표적화되었고, ZFNs을 사용하여 편집하였다[10]. 본 발명자들은 먼저 인간 HEK293T 세포 중의 sgRNA 및 스트렙토코쿠스 파이오제네스 Cas9 단백질의 인간-코돈 최적화된 버젼의 발현을 시험하였다. 160kDa Cas9 단백질은 HA 에피토프, 핵 국재화 신호(NLS), 및 Cas9의 C-말단에 부착된 그린 형광 단백질(GFP)을 포함하는 융합 단백질로서 발현되었다(도 29a). GFP-융합된 Cas9를 인코딩하는 벡터로 형질감염된 세포의 분석은 풍부한 Cas9 발현 및 핵 국재화(도 29b)를 나타냈다. 웨스턴 블랏팅은 Cas9 단백질이 이러한 세포로부터의 추출물에서 매우 순수하게 발현된다는 것을 확인하였다(도 29a). Cas9를 프로그래밍하기 위해, 본 발명자들은 표적 DNA 서열에 상보적인 5'-말단 20-뉴클레오타이드 서열 및 Cas9 결합에 필요한 42-뉴클레오타이드 3'-말단 줄기 루프 구조를 포함하는 sgRNA를 발현시켰다(도 29c). 이 3'-말단 서열은 시험관내에서 Cas9를 프로그래밍하기 위해 사용되었던 최소 줄기-루프 구조에 상응한다[8]. 이 sgRNA의 발현은 인간 U6 (RNA 폴리머라제 III) 프로모터[11]에 의해 구동되었다. U6 프로모터-구동된 sgRNA 플라스미드 발현 벡터로 형질감염된 세포로부터 추출된 RNA의 노던 블랏팅 분석은 sgRNA가 사실상 발현되고, 그들의 안정성은 Cas9의 존재로 증대된다는 것을 보여준다(도 29d).
도 29는 인간 세포에서 Cas9 및 가이드 RNA의 공-발현이 표적 유전자 좌에서 이중-가닥 DNA 파괴를 생성함을 입증한다. (A) 상단부; Cas9-HA-NLS-GFP 발현 작제물의 개략도. 하단부; Cas9 발현 플라스미드로 형질감염된 HEK293T 세포로부터 용해물을 항-HA 항체를 사용하여 웨스턴 블랏팅으로 분석하였다. (B) Cas9-HA-NLS-GFP를 발현시키는 HEK293T 세포의 형광 현미경검사. (C) 인간 CLTA 유전자 좌를 표적화하는 단일-가이드 RNA(sgRNA, 즉, 단일-분자 DNA-표적화 RNA)의 설계. 상단부; 인간 CLTA 유전자의 엑손 7 중의 sgRNA 표적 부위의 개략도. CLTA1 sgRNA의 가이드 세그먼트로 하이브리드화하는 표적 서열은 "CLTA1 sgRNA"로 나타낸다. GG 디-뉴클레오타이드 프로토스페이서 인접한 모티프(PAM)는 화살표로 마크된다. 블랙 라인은 대조군 ZFN 단백질의 DNA 결합 영역을 나타낸다. CLTA 개방 판독 프레임의 번역 정지 코돈은 참조를 위해 점선으로 마크한다. 중간; sgRNA 발현 작제물의 개략도. RNA는 U6 Pol III 프로모터 및 Pol III 전사 종결자 신호로서 작용하는 폴리(T) 관의 제어하에 발현된다. 하단부; Cas9에 의한 표적 DNA의 sgRNA-유도된 절단. sgRNA는 Cas9 결합에 필요한 42-nt 줄기-루프 구조가 따르는 20-nt 5'-말단 가이드 세그먼트로 이루어진다. 2개의 표적 DNA 가닥의 Cas9-매개된 절단은 표적 DNA의 풀기 및 sgRNA의 가이드 세그먼트과 표적 DNA 사이에 듀플렉스의 형성시 발생한다. 이것은 표적 DNA 중의 표적 서열의 PAM 모티프(사용된 Cas 9, 예를 들면, GG 디뉴클레오타이드, 상기 실시예 1 참조) 다운스트림의 존재에 의존한다. 표적 서열이 상부 다이아그램에 대해 역전된다는 것을 주시한다. (D) HEK239T 세포에서 sgRNA 발현의 노던 블랏 분석. (E) Cas9 및/또는 CLTA sgRNA를 발현시키는 HEK293T 세포로부터 단리된 게놈 DNA의 감독자 뉴클레아제 검정. CLTA 유전자 좌[10]를 표적화하기 위해 이전에 사용된 ZFN 작제물을 비-상동성 말단 결합에 의한 DSB-유도된 DNA 치유를 검출하기 위한 양성 대조군으로서 사용했다.
이어서, 본 발명자들은 부위 특이적 DSB가 Cas9-HA-NLS-mCherry 및 CLTA1 sgRNA로 형질감염된 HEK293T 세포에서 생성되는지를 조사했다. 이를 수행하기 위해, 본 발명자들은 감독자 뉴클레아제 검정[12]을 사용하여 DSB-유도된 NHEJ에 의한 불완전한 치유로부터 생성되는 유전자 좌에서 최소한의 삽입 및 결실에 대해 탐색했다. Cas9:sgRNA에 의해 표적화된 게놈 DNA의 영역은 PCR로 증폭시키고, 수득한 생성물은 변성되고 재어닐링된다. 재하이브리드화된 PCR 생성물은 불일치 인식 엔도뉴클레아제 Cel-1로 배양하고, 아크릴아미드 겔 상에서 분해하여 Cel-1 절단 밴드를 확인한다. NHEJ에 의한 DNA 치유가 전형적으로 DSB로 유도될 때, 감독자 검정에서 양성 신호는 게놈 DNA 절단이 발생하였음을 나타낸다. 이 검정을 사용하여, 본 발명자들은 CLTA1 sgRNA(도 29e)에 의해 표적화된 위치에서 CLTA 유전자 좌의 절단을 검출했다. CLTA 유전자 좌 중의 인접 부위를 표적화하는 ZFN의 쌍은 이러한 실험[10]에서 양성 대조군을 제공한다.
Cas9 또는 sgRNA 발현이 관찰된 게놈 편집 반응에서 제한 인자인지를 결정하기 위해, 형질감염된 세포로부터 제조된 용해물은 CLTA1 sgRNA로 표적화된 CLTA 유전자의 단편을 보유하는 플라스미드 DNA로 배양하였다. 플라스미드 DNA 절단은 감독자 검정 결과와 일치하여 Cas9-HA-NLS-GFP 발현 벡터 단독으로 형질감염된 세포로부터 제조된 용해물로 배양시 관찰되지 않았다. 그러나, 강력한 플라스미드 절단은 용해물이 시험관내 전사된 CLTA1 sgRNA(도 30a)로 보충될 경우에 검출되었다. 더욱이, Cas9 및 sgRNA 발현 벡터 둘 다로 형질감염된 세포로부터 제조된 용해물은 플라스미드 절단을 지지하는 반면, sgRNA-인코딩 벡터 단독으로 형질감염된 세포로부터의 용해물은 그렇지 않았다(도 30a). 이러한 결과는 인간 세포 중의 Cas9 기능을 위한 제한 인자가 sgRNA를 갖는 어셈블리일 수 있다는 것을 시사한다. 본 발명자들은 부가된 외인성 sgRNA의 존재 및 부재하에 이전과 같이 형질감염된 세포로부터의 용해물에서 플라스미드 절단을 직접적으로 분석하여 이 가능성을 시험하였다. 현저히, 외인성 sgRNA를 Cas9 및 sgRNA 발현 벡터 둘 다로 형질감염된 세포로부터의 용해물에 첨가될 경우, DNA 절단 활성의 실질적인 증가가 관찰되었다(도 30b). 이 결과는 HEK293T 세포에서 Cas9 기능을 위한 제한 인자가 Cas9에서 sgRNA의 발현 또는 Cas9로의 이의 부하이다는 것을 나타낸다.
도 30은 세포 용해물이 활성 Cas9:sgRNA를 함유하고, 부위 특이적인 DNA 절단을 지지한다는 것을 입증한다. (A) 좌측에서 명시된 플라스미드(들)로 형질감염된 세포로부터의 용해물을 PAM 및 CLTA1 sgRNA에 상보적인 표적 서열을 함유하는 플라스미드 DNA로 배양하고; 명시될 경우, 반응물을 10pmol의 시험관내 전사된 CLTA1 sgRNA로 보충시키고; XhoI에 의한 2차 절단은 Cas9-매개된 절단을 나타내는 약 2230 및 약 3100bp 단편의 단편을 생성하였다. ZFN 발현 작제물로 형질감염된 세포로부터의 용해물을 사용하는 대조군 반응은 CLTA1 표적 부위에 대해 ZFN 표적 부위의 오프셋을 반영하는 약간 상이한 크기의 단편을 나타낸다. (B) Cas9-GFP 발현 플라스미드 및, 명시될 경우, CLTA1 sgRNA 발현 플라스미드로 형질감염된 세포로부터의 용해물을 시험관내-전사된 CLTA1 sgRNA의 부재 또는 존재하에 (A)에서와 같이 표적 플라스미드 DNA로 배양하였다.
살아 있는 세포에서 Cas9:sgRNA 어셈블리를 강화하는 수단으로서, 본 발명자들은 이어서 가이드 RNA의 추정된 Cas9-결합 영역을 확장하는 효과를 시험하였다. CLTA1 sgRNA의 두 개의 새로운 버젼을 crRNA 및 tracrRNA 사이의 염기-짝짓기 상호작용을 모방하는 나선에 추가의 6개 또는 12개 염기쌍을 포함하도록 설계하였다(도 31a). 추가로, 가이드 RNA의 3'-말단은 에스. 파이오제네스 tracrRNA의 천연 서열을 기본으로 하는 5개 뉴클레오타이드로 확장시켰다[9]. U6 또는 H1 Pol III 프로모터의 제어하에 이러한 3' 확장된 sgRNA를 인코딩하는 벡터를 Cas9-HA-NLS-GFP 발현 벡터와 함께 세포로 형질감염시키고, 부위 특이적인 게놈 절단은 감독자 검정을 사용하여 시험하였다(도 31b). 결과는 절단이 Cas9 및 CLTA1 sgRNA 둘 다를 필요로했다는 것을 확인하였지만, Cas9 또는 sgRNA이 단독으로 발현될 경우에는 발생하지 않았다. 더욱이, 본 발명자들은 Cel-1 뉴클레아제 절단에 의해 검출된 바와 같이 실질적으로 증가된 NHEJ의 빈도를 관찰하였지만, 대조군 ZFN 쌍으로 수득된 NHEJ 돌연변이유발 빈도는 크게 변하지 않았다.
도 31은 sgRNA 작제물의 3' 확대는 부위 특이적인 NHEJ-매개된 돌연변이유발을 강화한다는 것을 증명한다. (A) CLTA1 sgRNA 발현의 작제물(상단부)은 원래 Cas9-결합 서열 (v1.0), 또는 4개 염기쌍(v2.1) 또는 10개 염기쌍(v2.2)으로 확장된 dsRNA 듀플렉스를 함유하는 전사체를 생성하도록 설계되었다. (B) Cas9 및/또는 CLTA sgRNA v1.0, v2.1 또는 v2.2를 발현시키는 HEK293T 세포로부터 단리된 게놈 DNA의 감독자 뉴클레아제 검정. CLTA 유전자 좌[10]를 표적화하는데 이전에 사용된 ZFN 작제물을 비-상동성 말단 결합으로 DSB-유도된 DNA 치유를 검출하기 위한 양성 대조군으로서 사용되었다.
따라서, 결과는 다양한 게놈 편집 적용을 위한 손쉬운 분자 도구로서 Cas9를 구현시키기 위한 뼈대를 제공한다. 이러한 시스템의 강력한 특징은 단일 유전자 좌에서 표적화 효율을 증가시키기 위해 또는 다수의 유전자 좌를 동시에 표적화하는 수단으로써 동일 세포에서 다중 sgRNA로 Cas9를 프로그래밍하는 가능성이다. 이러한 전략은 다유전자적 질환 모델의 개발과 같은 대규모 연구 노력 및 게놈 규모 실험에서 광범위한 적용을 찾을 수 있다.
실시예 3: 유형 II CRISPR-Cas 면역력 시스템의 tracrRNA 및 Cas9 패밀리
본 발명자들은 유형 II 시스템의 홀마크 단백질인 Cas9에 상동성인 서열을 스크리닝하여 공공연하게 이용가능한 박테리아 게놈에 현재 존재하는 모든 추정 유형 II CRISPR-Cas 유전자 좌를 탐색했다. 본 발명자들은 확인된 Cas9 오쏘로그의 다중 서열 정렬로부터 계통수를 구성했다. 관련된 유형 II 시스템의 cas 오페론의 유전자 구성 및 CRISPR 반복체 길이를 상이한 Cas9 서브클러스터로 분석했다. 유형 II 유전자 좌의 하위 분류가 제안되었고, 추가로 75 대표적인 Cas9 오쏘로그의 선택을 기준으로 하는 서브그룹으로 분할하였다. 이어서, 본 발명자들은 주로 CRISPR 반복 서열을 검색하고, 선택된 유형 II 유전자 좌의 CRISPR 배열 및 cas 유전자 내에서 또는 부근에서 항-반복체를 스크리닝하여 tracrRNA 서열을 예상했다. 서열 및 선택된 tracrRNA 오쏘로그의 예상된 구조의 비교 분석을 수행하였다. 마지막으로, 본 발명자들은 5개 박테리아 종으로부터 tracrRNA 및 crRNA의 발현 및 처리 프로파일을 결정했다.
물질 및 방법
균주 및 배양 조건
하기의 배지를 사용하여 박테리아를 플레이트 상에서 성장시켰다: 에스. 뮤탄스 (UA159)의 경우 3% 양 혈액이 보충된 TSA(트립티카제 대두 한천, 트립티카제TM 대두 한천 (TSA II) BD BBL, Becton Dickinson), 및 엘. 이노쿠아(Clip11262)의 경우 BHI(뇌 심장 주입, BD BactoTM 뇌 심장 주입, Becton Dickinson) 한천. 액체 배양액 중에서 배양하면, 0.2% 효모 추출물 (ServabacterR)이 보충된 THY 배지(토드 휴이트 액체배지 (THB, Bacto, Becton Dickinson)를 에스. 뮤탄스를 위해, 엘. 이노쿠아를 위해 BHI 액체배지를, 엔. 메닌자이티디스 (A Z2491)를 위해 1% 비타민-혼합물 VX(Difco, Becton Dickinson)를 포함하는 BHI 액체 배지, 씨. 제주니 (NCTC 11168; ATCC 700819)를 위해 1% 비타민-혼합물 VX를 포함하는 MH (무엘러 힌톤 액체배지, Oxoid) 액체배지를, 에프. 노비시다 (U112)를 위해 TSB(트립신 간장 액체배지, BD BBLTM 트립티카제 간장 액체배지)를 사용했다. 에스. 뮤탄스를 진탕 없이 37℃, 5% CO2에서 배양하였다. 엘. 이노쿠아, 엔. 메닌자이티디스 및 에프. 노비시다의 균주를 진탕시키면서 37℃에서 호기적으로 성장시켰다. 씨. 제주니를 캠피건(옥소이드) 대기를 사용하여 미호기적 조건에서 37℃에서 성장시켰다. 마이크로플레이트 판독기(BioTek PowerWaveTM)를 사용하여 규칙적인 시간 간격으로 620nm에서 배양물의 광학 밀도(OD620 nm)를 측정함으로써 박테리아 세포 성장이 따랐다.
박테리아 작은 RNA 라이브러리의 서열분석
씨. 제주니 NCTC 11168 (ATCC 700819), 에프. 노비시다 U112, 엘. 이노쿠아 Clip11262, 엔. 메닌자이티디스 A Z2491 및 에스. 뮤탄스 UA159를 중간-대수 성장 단계까지 배양하고, 총 RNA를 TRIzol(시그마-알드리치)로 추출시켰다. 각 균주로부터 10㎍의 총 RNA를 TURBOR DNase(Ambion)로 처리하여 임의의 잔류 게놈 DNA을 제거하였다. 리보솜 RNA은 제조업자의 지침에 따라서 그람-양성 또는 그람-음성 박테리아(Epicentre)에 대해 Ribo-제로TM rRNA 제거 Kits®을 사용하여 제거하였다. RNA 클린 & 콘센트레이터TM-5 kit(Zymo Research)로 정제 후, 라이브러리를 제조업자 지침에 따라서 ScriptMinerTM Small RNA-Seq 라이브러리 제조 키트(Multiplex, Illumina® 양립가능)를 사용하여 제조하였다. RNA를 담배 산 파이로포스파타제(TAP)(Epicentre)로 처리하였다. RNA 클린 & 콘센트레이터TM-5(Zymo Research)로부터의 컬럼을 후속적인 RNA 정제용으로 사용하였고, Phusion® 고충실도 DNA 폴리머라제(New England Biolabs)를 PCR 증폭용으로 사용하였다. 특이적인 사용자규정된 바코드를 각 라이브러리(RNA-Seq 바코드 프라이머(Illumina®-양립가능) Epicentre)에 부가하였고, 샘플을 호주 비엔나 비엔나 바이오센터의 차세대 서열분석(CSF NGS Unit; "csf."에 이어 "ac.at"에서 웹상에서) 설비에서 서열분석하였다(일루미나 단일 말단 서열분석).
tracrRNA 및 crRNA 서열분석 데이타의 분석
RNA 서열분석 판독치를 일루미나2밤 도구를 사용하여 분할하고, (i) 일루미나 어댑터 서열 (cutadapt 1.0)의 제거 및 (ii) 3' 말단에서 15 nt의 제거로 손질하여 판독 품질을 향상시켰다. 15nt보다 짧은 판독치의 제거 후, cDNA 판독치를 2개의 불일치를 허용함으로써 보우타이를 사용하여 그들의 각각의 게놈으로 정렬시켰다: 씨. 제주니 (GenBank: NC_002163), 에프. 노비시다 (GenBank: NC_008601), 엔. 메닌자이티디스 (GenBank: NC_003116), 엘. 이노쿠아 (GenBank: NC_003212) 및 에스. 뮤탄스 (GenBank: NC_004350). 판독치의 적용범위는 BEDTools-버전-2.15.0를 사용하여 두 DNA 가닥에 대해 별도로 각 뉴클레오타이드 위치에서 계산하였다. 백만당 판독치(rpm)의 적용범위를 함유하는 정상화된 위글 파일을 생성하고, 통합 유전체학 뷰어(IGV) 도구("www."에 이어 "broadinstitute.org/igv/")(도 36)를 사용하여 가시화하였다. SAMTools flagstat80을 사용하여 맵핑된 판독치의 비율을 총 맵핑된 씨. 제주니에 대한 9914184 판독치, 에프. 노비시다에 대한 48205 판독치, 엔. 메닌자이티디스에 대한 13110087 판독치, 엘. 이노쿠아에 대한 161865 판독치 및 에스. 뮤탄스에 대한 1542239 판독치에 대해 계산하였다. 각 단일 뉴클레오타이드 위치에서 개시(5') 및 말단(3') 판독치의 수를 함유하는 파일을 생성하고, IGV로 가시화하였다. 각 tracrRNA 오쏘로그 및 crRNA를 위해, 검색된 판독치의 총수를 SAMtools을 사용하여 계산하였다.
Cas9 서열 분석, 다중 서열 정렬 및 가이드 트리 구성
위치-특이적 반복된(PSI)-BLAST 프로그램을 NCBI 비중복 데이타베이스로 Cas9 패밀리의 동족체를 검색하는데 사용하였다. 800 아미노산보다 짧은 서열은 폐기했다. 길이 적용범위 컷오프 0.8 및 스코어 적용범위 역치(정렬 길이로 분할된 비트 스코어) 0.8로 설정된 BLASTClust 프로그램을 잔여 서열(도 38)을 클러스터링하는데 사용하였다. 이 절차는 78개 클러스터를 생산했다(그들 중 48개는 단지 하나의 서열로 제시되었다). 하나 (또는 드물게 소수의 대표)를 각 클러스터로부터 선택하고, 이러한 서열을 위한 다중 정렬을 디폴트 파라미터를 갖는 근육 프로그램을 사용한 다음, PSI-BLAST 및 HHpred 프로그램을 사용하여 수득된 국소 정렬을 기초로 하여 수동 보정으로 구성하였다. 몇몇 추가 서열은 정렬되지 않았고, 또한 최종 서열에서 제외되었다. 272개 유익한 정보를 주는 위치를 갖는 자신있게 정렬된 블록은 디폴트 파라미터: JTT 진화론적 모델, 20 속도 카테고리를 갖는 별개의 감마 모델을 갖는 FastTree 프로그램을 사용하여 최대 가능성 트리 재구성을 위해 사용했다. 동일한 프로그램을 부트스트랩 값을 계산하는데 사용하였다.
도 38은 BLASTclust 클러스터링 프로그램에 따라 그룹화된 서열을 묘사한다. 800 아미노산보다 긴 서열만을 BLASTclust 분석(참조: 물질 및 방법)을 위해 선택했다. cas9 오쏘로그 유전자를 보유하는 대표적인 균주를 사용했다. 몇몇 서열은 클러스터화되지 않았지만, 그들 바로 근처에서 보존된 모티프 및/또는 기타 csa 유전자의 존재에 기인하여 Cas9 서열로서 증명되었다.
CRISPR-Cas 유전자 좌의 분석
CRISPR 반복 서열을 CRISPRdb 데이타베이스로부터 검색하거나 CRISPRFinder 도구(참조: Grissa I 등, BMC Bioinformatics 2007; 8:172; Grissa I 등, Nucleic Acids Res 2007)를 사용하여 예상했다. cas 유전자는 BLASTp 알고리즘을 사용하여 확인하고/하거나 KEGG 데이타베이스("www."에 kegg.jp/가 따르는 웹 상에서)로 증명하였다.
tracrRNA 오쏘로그의 인실리코 예측 및 분석
추정 항반복체는 15개 이하의 불일치를 허용하는 각각의 게놈의 두 가닥 상의 반복체-스페이서 어레이에 속하지 않은 추가의 변성된 반복체 서열을 스크리닝하여 벡터 NTIR 소프트웨어(Invitrogen)를 사용하여 확인하였다. 전사 프로모터 및 로우-독립적인 종결자는 각각 BDGP 신경 네트워크 프로모터 예측 프로그램("www."에 이어 fruitfly.org/seq_tools/promoter.html이 따름) 및 TransTermHP 소프트웨어를 사용하여 예상하였다. 다중 서열 정렬은 디폴트 파라미터를 갖는 근육 프로그램을 사용하여 수행하였다. 정렬은 비엔타 RNA 패키지 2.0의 RNAalifold 알고리즘을 사용하여 보존된 구조 모티프의 존재에 대해 분석하였다.
결과
유형 II CRISPR-Cas 시스템은 박테리아에서 일반적이다.
tracrRNA-인코딩 DNA 및 반복체-스페이서 어레이 이외에, 유형 II CRISPR-Cas 유전자 좌는 전형적으로 오페론(도 32a-b)으로 조직된 3 내지 4개의 cas 유전자로 구성된다. Cas9는 유형 II에 특징적인 서명 단백질이고, 발현 및 간섭 단계에 관여한다. Cas1 및 Cas2는 모든 CRISPR-Cas 시스템에 의해 공유되고, 스페이서 취득에 연루되는 코어 단백질이다. Csn2 및 Cas4는 단지 유형 II의 서브세트로 존재하고, 적응에서 역할을 하는 것으로 제안되었다. tracrRNA를 함유하는 유형 II CRISPR-Cas 유전자 좌의 최대 수를 검색하기 위해, 본 발명자들은 먼저 이미 주석이 달린 Cas9 단백질에 상동성인 서열을 위해 공공연하게 이용가능한 게놈을 스크리닝하였다. 235 Cas9 오쏘로그는 203 박테리아 종에서 확인되었다. 모든 검색된 cas9 오쏘로그를 대표하는 75 다양한 서열의 세트를 추가 분석용으로 선택하였다(도 32, 도 38, 및 물질 및 방법).
도 32 (A) 다양한 유기체로부터 대표적인 Cas9 서열의 계통수 뿐만 아니라 (B) 대표적인 Cas9 유전자 좌위 구조를 묘사한다. 각각의 노드에 대해 계산된 부트스트랩 값이 명시된다. 동일한 색상 분지는 유사한 Cas9 오쏘로그의 선택된 서브클러스터를 나타낸다. 뉴클레오타이드 중의 CRISPR 반복체 길이, 아미노산(aa)의 평균 Cas9 단백질 크기 및 공유서열 유전자 좌위 구조가 각 서브클러스터에 대해 제시된다. *- gi|116628213 **- gi|116627542 †- gi|34557790 ‡ gi|34557932. 유형 II-A는 cas9- csx12, cas1, cas2, cas4로 특성화된다. 유형 II-B는 cas9, cas1, cas2에 이어, csn2 변이체로 특성화된다. 유형 II-C는 보존된 cas9, cas1, cas2 오페론(또한 도 38을 참조한다)에 의해 특성화된다.
다음으로, 본 발명자들은 선택된 Cas9 오쏘로그의 다중 서열 정렬을 수행하였다. 비교 분석은 아미노산 조성 및 단백질 크기에서 높은 다양성을 나타냈다. Cas9 오쏘로그는 단지 몇몇 동일한 아미노산을 공유하고, 모든 검색된 서열은 중앙 HNH 엔도뉴클레아제 도메인 및 분할된 RuvC/RNaseH 도메인과 동일한 도메인 구조를 갖는다. Cas9 단백질의 길이는 전형적인 크기 약 1100 또는 약 1400 아미노산과 함께 984(캠필로박터 제주니) 내지 1629(프란시셀라 노비시다) 아미노산의 범위이다. 특히 도메인간 영역의 길이에서 Cas9 서열의 높은 다양성에 기인하여, 본 발명자들은 분석된 서열의 계통수를 재작제하기 위해 제조된 정렬의 단지 충분히 정렬된 유익한 정보를 주는 위치를 선택했다(도 32 및 물질 및 방법). Cas9 오쏘로그는 몇몇 특이 서열을 갖는 3개의 주요 단일 종족 클러스터로 그룹화된다. Cas9 트리의 관찰된 위상기하학은 유형 II 유전자 좌의 현재 분류와 잘 일치하고, 이전에 규정된 유형 II-A 및 유형 II-B는 별도의 단일 종족 클러스터를 형성한다. 클러스터를 추가로 특성화하기 위해, 본 발명자들은 모든 나열된 균주의 cas 오페론 조성물 및 CRISPR 반복 서열을 상세히 시험하였다.
Cas9 서브클러스터링은 유형 II CRISPR-Cas 유전자 좌위 구조의 다양성을 반영한다
선택된 유형 II 유전자 좌의 보다 깊은 분석은, Cas9 오쏘로그 서열의 클러스터링이 CRISPR 반복체 길이의 다양성과 서로 관련있음을 나타냈다. 유형 II CRISPR-Cas 시스템의 대부분은, 반복체 길이가 2개의 Cas9 트리 서브클러스터에 대한 약간의 변형과 함께 36 뉴클레오타이드(nt)이다. 이전에 Csx12로 명명된 긴 Cas9 오쏘로그를 인코딩하는 유전자 좌를 포함하는 유형 II-A 클러스터(도 32)에서, CRISPR 반복체는 길이 37nt이다. 박테로이데테스 문(도 32)에 속하는 박테리아로부터의 서열로 구성된 작은 서브클러스터는 크기가 48nt 이하인 매우 긴 CRISPR 반복체를 특징으로 한다. 더욱이, 본 발명자들은 Cas9 서열의 서브클러스터링이 도 32에 묘사된 바와 같이 별개의 cas 오페론 구조와 서로 관련있다는 것을 주시했다. 제 3 주요 클러스터(도 32) 및 특이 유전자 좌(도 32)는 이후에 논의되는 일부 불완전한 유전자 좌를 제외하고 cas9, cas1 및 cas2 유전자로 구성된 최소 오페론으로 주로 이루어진다. 2개의 제 1 주요 클러스터의 모든 기타 유전자 좌는 제4 유전자, 주로 유형 II-A에 특이적인 cas4 또는 유형 II-B에 특이적인 csn2-형과 연관된다(도 32). 본 발명자들은 유형 II-B 에스. 파이오제네스 CRISPR01 및 에스. 서모필러스 CRISPR3과 유사한 유전자 좌 내에서 Csn2 단백질의 짧은 변이체, Csn2a를 인코딩하는 유전자를 확인하였다(도 32). Csn2의 보다 긴 변이체, Csn2b는 유형 II-B 에스. 서모필러스 CRISPR1과 유사한 유전자 좌와 연관되는 것으로 밝혀졌다(도 32). 흥미롭게도, 본 발명자들은 이전에 기재된 Csn2 변이체에 대한 명백한 서열 유사성을 갖지 않는 단백질을 인코딩하는 추가의 추정 cas 유전자를 확인하였다. 이러한 무특성화 단백질 중의 하나는 배타적으로 마이코플라스마 종의 유형 II-B 유전자 좌에 연관된다(도 32 및 도 33). 2개의 다른 것은 스타필로코쿠스 종의 유형 II-B 유전자 좌에서 인코딩되는 것으로 밝혀졌다(도 33). 모든 경우에, cas 오페론 구조 다양성은 따라서 Cas9 서열의 서브클러스터링과 일치한다. 이들 특성은 3개의 주요 명확한 단일 종족 클러스터로 분할된 Cas9 트리의 일반적인 위상기하학과 함께 유형 II CRISPR-Cas 시스템을 3개의 서브유형으로의 새로운 추가 분열을 제안하도록 한다. 도 32에 묘사된 바와 같이, 유형 II-A는 Csx12-형 Cas9 및 Cas4와 연관되고, 유형 II-B는 Csn2-형과 연관되고, 유형 II-C는 단지 cas9, cas1 및 cas2 유전자의 최소 세트를 함유한다.
도 33은 선택된 박테리아 종으로부터의 유형 II CRISPR-Cas의 구조를 묘사한다. 수직 막대는 동일 트리 서브클러스터에 속하는 Cas9 오쏘로그를 코딩하는 유전자 좌를 그룹화한다(도 32와 비교). 수평 블랙 막대, 리더 서열; 블랙 직사각형 및 다이아몬드, 반복체-스페이서 어레이. 예상된 항-반복체는 추정 tracrRNA 오쏘로그 전사의 방향을 나타내는 화살표로 나타낸다. 실험적으로 증명되지 않은 유전자 좌의 경우, CRISPR 반복체-스페이서 어레이가 cas 오페론과 동일한 가닥으로부터 전사되는 것으로 본원에서 간주된다. 추정 tracrRNA 오쏘로그의 전사 방향이 따라서 명시된다.
신규한 tracrRNA 오쏘로그의 인실리코 예측
75 대표적인 Cas9 오쏘로그를 기반으로 하여 초기에 선택된 유형 II 유전자 좌를 추정 tracrRNA 오쏘로그의 존재에 대해 스크리닝하였다. 제한된 수의 tracrRNA 서열에 대해 수행된 본 이전 분석은 CRISPR-Cas 유전자 좌 내에서 tracrRNA 서열도 그들의 국재화도 보존되지 않는 것으로 보인다고 나타냈다. 그러나, 상기에서 언급된 바와 같이, tracrRNA는 Cas9의 존재하에 RNase III으로 절단되는 tracrRNA:전crRNA 반복체 듀플렉스를 형성하기 위해 전-crRNA 반복체 각각과 염기 짝짓기할 수 있는 항-반복체 서열을 특징으로 한다. 신규한 tracrRNA를 예상하기 위해, 본 발명자들은 이 특징을 이용하고 다음 작업의 흐름을 사용했다: (i) CRISPR-Cas 유전자 좌 내에서 잠재적인 항-반복체(CRISPR 반복체와 서열 염기 짝짓기)를 스크리닝하고, (ii) 유전자간 영역에 위치한 항-반복체를 선택하고, (iii) CRISPR 항-반복체:반복체 염기 짝짓기를 실증하고, (iv) 확인된 tracrRNA에 관련된 프로모터 및 로우-독립적인 전사 종결자를 예상한다.
추정 항-반복체를 스크리닝하기 위해, 본 발명자들은 CRISPRdb 데이타베이스로부터 반복 서열을 회수하거나, 정보가 이용가능하지 않을 경우, 본 발명자들은 CRISPRfinder 소프트웨어를 사용하여 반복체 서열을 예상했다. 본 이전 연구에서, 본 발명자들은 반복체-스페이서 어레이의 전사 방향이 유전자 좌 중에서 변화된 cas 오페론의 것과 비교된다는 것을 실험적으로 보여주었다. 여기서, RNA 서열분석 분석은 이 관찰을 확인하였다. 분석된 유전자 좌 중의 일부에서, 즉 에프. 노비시다, 엔. 메닌자이티디스 및 씨. 제주니에서, 반복-스페이서 어레이는 cas 오페론의 반대 방향으로 전사되고(참조: 단락 '깊은 RNA 서열분석은 신규한 tracrRNA 오쏘로그의 발현을 실증한다' 및 도 33 및 34), 반면에 에스. 파이오제네스, 에스. 뮤탄스, 에스. 서모필러스 및 엘. 이노쿠아에서, 상기 어레이 및 상기 cas 오페론은 동일한 방향으로 전사된다. 현재까지 이용가능한 유일한 유형 II 반복체-스페이서 어레이 발현 데이타가 있다. 기타 반복체-스페이서 어레이의 전사 방향을 예상하기 위해, 본 발명자들은 어레이의 최종 반복체가 보통 돌연변이됨에 따르는 이전 관찰을 고려했다. 이 관찰은 현재 스페이서 취득 모델과 일치하고, 여기서 전형적으로 어레이의 제 1 반복체는 적응 단계 동안 스페이서 서열의 삽입시 중복된다. 37 반복체 스페이서 어레이의 경우, 본 발명자들은 어레이의 추정 말단에서 돌연변이된 반복체를 확인할 수 있었다. 본 발명자들은 엔. 메닌자이티디스 및 씨. 제주니 반복체-스페이서 어레이의 예상된 전사 배향이 실험적으로 결정된 배향(RNA 서열분석 및 노던 블랏 분석)과 반대일 것이다는 것을 관찰했다. 예상된 배향이 클러스터와 일치하지 않고 대부분의 경우 본 발명자들이 어레이의 양 말단에서 잠재적인 프로모터를 검출할 수 있기 때문에, 본 발명자들은 반복체-스페이서 어레이의 전사가 다르게 실증되지 않는다면, cas 오페론의 전사와 동일한 방향일 것으로 간주했다.
도 34는 선택된 유형 II CRISPR Cas 시스템 중에서 공-발현하는 tracrRNA 및 전-crRNA를 묘사한다. 증명된 tracrRNA 및 전-crRNA 전사 위치 및 방향을 갖는 CRISPR 유전자 좌위 구조가 제시된다. 상단부 서열, 전-crRNA 반복체; 하단부 서열, crRNA 반복체와 tracrRNA 서열 염기 짝짓기. RNA 서열분석으로 나타낸 추정 RNA 처리 부위는 화살촉으로 나타낸다. 각 유전자 좌의 경우, 화살촉 크기는 회수된 5' 및 3' 말단의 상대적인 양을 나타낸다(또한, 도 37 참조).
도 37은 좌표(목적하는 영역) 및 상응하는 cDNA 서열(5' 내지 3')을 포함하여 연구되는 박테리아 종에 대한 서열분석으로 회수된 모든 tracrRNA 오쏘로그 및 성숙한 crRNA를 나열한다. 화살표는 전사 방향(가닥)을 나타낸다. cDNA 판독치의 수(SAMtools을 사용하여 계산됨), 적용범위 수(맵핑된 판독치의 백분율) 및 각 전사체와 관련된 두드러진 말단이 명시된다. 각 전사체의 5' 및 3' 말단 주위의 각 뉴클레오타이드 위치에서 개시하거나 정지하는 판독치의 수를 나타낸다. 각 crRNA 성숙한 형태의 크기가 명시된다. 각 crRNA 종에 할당된 수는 CRISPRdb에 따라서 전-crRNA 중의 스페이서 서열 위치에 상응한다. 각 tracrRNA 종에 할당된 수는 동일한 전사체의 상이한 형태에 상응한다.
이어서, 본 발명자들은 15개 이하의 불일치를 허용하는 반복체-스페이서 어레이에 속하지 않는 가능한 반복체 서열에 대해 두 가닥 상의 업스트림 및 다운스트림 1kb에 위치된 서열을 포함하는 선택된 CRISPR-Cas 유전자 좌를 스크리닝하였다. 평균적으로, 본 발명자들은 tracrRNA 오쏘로그의 항-반복체에 상응하는 유전자 좌당 1 내지 3개의 변성된 반복체 서열을 밝혀냈고, 유전자 간 영역 내에 위치된 서열을 선택했다. 추정 항-반복체는 4개의 전형적인 국재화: cas9 유전자의 업스트림, cas9 및 cas1 사이의 영역, 및 반복체-스페이서 어레이의 업스트림 또는 다운스트림에서 발견되었다(도 33). 모든 회수된 서열에 대해, 본 발명자들은 가능한 RNA:RNA 상호작용을 예상하고, 특히 RNase III 처리를 위한 최적의 이중-가닥 구조를 형성하는 보다 길고 완전한 상보성 영역을 갖는 후보에 집중시킴으로써 반복체와 항-반복체(도 44) 사이에 형성된 염기 짝짓기의 정도를 확인했다. 항-반복체 측면에 위치한 프로모터 및 전사 종결자를 예상하기 위해, 본 발명자들은 추정 전사 개시 및 종결 부위를 본 이전 관찰26을 기준으로 하여, 각각 항-반복체 서열의 최대 200nt 업스트림 및 100nt 다운스트림에 위치된 영역 내에 포함되도록 설정했다26. 상기에서 언급된 바와 같이, 유형 II 시스템의 대부분의 반복체-스페이서 어레이의 전사 방향에 대한 실험 정보는 부족하다. 인실리코 프로모터 예측 알고리즘은 흔히 두 가닥으로부터 반복체-스페이서 어레이의 전사를 유도할 수 있는 추정 프로모터에 거짓 양성 결과 및 점을 제공한다. 몇몇 경우에, 본 발명자들은 씨. 제주니 유전자 좌로 예시된 바와 같이(참조: 단락 '깊은 RNA 서열분석이 신규한 tracrRNA 오쏘로그의 발현을 확인한다'), tracrRNA 오쏘로그 발현이 실험적으로 확인될 수 있지만, 본 발명자들은 전사 종결자를 예상할 수 없었다. 본 발명자들은 프로모터 및 전사 종결자 예상을 단지 상기한 가이드라인의 보완적이지만 필수적이지 않은 단계로서 간주하도록 제안한다.
도 44는 선택된 박테리아 종에서 예상된 전-crRNA 반복체:tracrRNA 항-반복체 염기 짝짓기를 묘사한다. bCRISPR 유전자 좌는 유형 II(Nmeni/CASS4) CRISPR-Cas 시스템에 속한다. 명명법은 CRISPR 데이타베이스(CRISPRdb)에 따른다. 에스. 서모필러스 LMD-9 및 더블유. 석시노게네스가 두 유형 II 유전자 좌를 함유한다는 것을 주시한다. c상부 서열, 전-crRNA 반복체 공유 서열(5' 내지 3'); 하부 서열, 반복체를 어닐링하는 tracrRNA 동족체 서열(항-반복체; 3' 내지 5'). 제공된 반복체 서열은 CRISPR 반복체-스페이서 어레이가 cas 오페론과 동일한 가닥으로부터 전사된다는 가정에 기초한다는 것을 주시한다. 이 연구에서 실험적으로 확인된 서열의 경우, RNA 서열분석 데이타는 염기 짝짓기를 결정하기 위해 고려되었다. 도 33 참조. d두 개의 가능한 항-반복체를 에프. 툴라렌시스 subsp. 노비시다, 더블유. 석시노게네스 및 감마 프로테오박테리움 HTCC5015 유형 II-A 유전자 좌에서 확인했다. 상부 서열 짝짓기, 추정 리더 서열 내의 항-반복체; 하부 서열 짝짓기, 반복 스페이서 어레이의 항-반복체 다운스트림. 도 33 참조. e두 개의 가능한 항-반복체는 S. 워즈워텐시스(wadsworthensis) 유형 II-A 유전자 좌에서 확인되었다. 상부 서열 짝짓기, 항-반복체; 하부 서열 짝짓기, 추정 리더 서열 내의 항-반복체. 도 33 참조. f두 개의 가능한 항-반복체는 엘. 가쎄리 유형 II-B 유전자 좌에서 확인되었다. 상부 서열 짝짓기, Cas9의 항-반복체 업스트림; 하부 서열 짝짓기, cas9 및 cas1 유전자 사이의 항-반복체. 도 33 참조. g두 개의 가능한 항-반복체는 씨. 제주니 유형 II-C 유전자 좌에서 확인되었다. 상부 서열 짝짓기, cas9의 항-반복체 업스트림; 하부 서열 짝짓기, 반복체-스페이서 어레이의 항-반복체 다운스트림. 도 33 참조. h두 개의 가능한 항-반복체는 R. 루브럼 유형 II-C 유전자 좌에서 확인되었다. 상부 서열 짝짓기, 반복-스페이서 어레이의 항반복체 다운스트림; 하부 서열 짝짓기, cas1의 항-반복체 업스트림. 도 33 참조.
tracrRNA 오쏘로그의 과다
본 발명자들은 조기에 선택된 75개 유전자 좌 중 56개에 대해 추정 tracrRNA 오쏘로그를 예상했다. 예상 결과는 도 33에 묘사된다. 이미 언급된 바와 같이, 이 도면에서 명시된 tracrRNA 전사 방향은 가정적이고, 반복체-스페이서 어레이의 지시된 전사 방향을 기반으로 한다. 이전에 언급된 바와 같이, 추정 tracrRNA 오쏘로그를 인코딩하는 서열은 cas 오페론 업스트림, 내부 및 다운스트림 뿐만 아니라 유형 II-A 유전자 좌(도 33)에서 통상 발견되는 추정 리더 서열을 포함하는 반복체 스페이서 어레이의 다운스트림에서 확인되었다. 그러나, 본 발명자들은 CRISPR-Cas 유전자 좌 내의 유사한 국재화의 항-반복체가 상이한 방향(예를 들면, 락토바실러스 람노서스 및 유박테륨 렉탈레 또는 마이코플라스마 모빌레 및 에스. 파이오제네스 또는 엔. 메닌자이티디스를 비교할 때 관찰된)으로 전사될 수 있다는 것을 관찰했다(도 33). 현저히, Cas9 가이드 트리의 동일한 서브클러스터 내에서 그룹화된 유전자 좌는 tracrRNA-인코딩 유전자의 위치와 관련하여 공통 구조를 공유한다. 본 발명자들은 유형 II-A 유전자 좌에서 반복체-스페이서 어레이 주위에서 및 대개 유형 II-B의 3개의 명확한 서브클러스터에서 cas9와 cas1 사이에 위치된 추정 tracrRNA에 대한 몇몇 주목할 만한 예외와 함께, 유형 II-B 및 II-C에서 Cas9 유전자의 업스트림에서 항-반복체를 확인했다.
일부 유형 II CRISPR-Cas 유전자 좌는 불완전한 반복체-스페이서 어레이 및/또는 tracrRNA 오쏘로그를 갖는다
6개 유형 II 유전자 좌(푸소박테리움 뉴클레아툼, 아미노모나스 포시보란스, 헬리코박터 무스텔라에, 아조스피릴룸 sp., 푸레보텔라 루미니콜라 및 악케르만시아 뮤시니필라)에 대해, 본 발명자들은 반복체 서열에 대한 약한 염기 짝짓기를 갖거나 개방 해독틀 내에 위치된 잠재적인 항-반복체를 확인했다. 현저히, 이러한 유전자 좌에서, A. 포시보란스에서 추정 ATPase를 인코딩하는 개방 해독틀 내의 약한 항-반복체, 아조스피릴룸 sp. B510에서 cas9 유전자의 제 1 100nt 내의 강한 항-반복체 및 A. 무시니필라에서 cas9 및 cas1 둘 다와 중첩하는 강한 항-반복체가 확인되었다(도 33). 12개의 추가의 유전자 좌(펩토니필루스 두에르데니, 코프로코쿠스 카투스, 악시다미노코쿠스 인테스티니, 카테니박테리움 미쓰오카이, 스타필로코쿠스 슈딘테르메디우스, 일료박터 폴리트로푸스, 에루시미크로비움 미누툼, 박테로이드 프라길리스, 악시도테르무스 셀루로라이티쿠스, 코라이네박테리움 디프테리아에, 비피더박테리움 롱굼 및 비피더박테리움 덴티움)의 경우, 본 발명자들은 임의의 추정 항-반복체를 검출할 수 없었다. 이러한 CRISPR-Cas 유전자 좌에서 전-crRNA 발현 및 처리에 대한 이용가능한 정보는 없다. 따라서, 명백하게 규정된 tracrRNA 오쏘로그의 부재하에 유형 II 시스템의 기능성은 처리되도록 잔류한다. 7개의 분석된 유전자 좌의 경우, 본 발명자들은 임의의 반복체 스페이서 어레이(파라수테렐라 에크리멘티호미니스, 바실러스 세레우스, 루미노코쿠스 알부스, 로도슈도모나스 팔루스트리스, 니트로박터 햄부르겐시스, 브래디히조비움 sp. 및 프레보텔라 미칸스)(도 33)를 확인할 수 없었고, 이들 중 3개(브래디히조비움 sp. BTAi1, N. 햅부르겐시스 및 B. 세레우스)에서, 본 발명자들은 부근에 기타 cas 유전자를 갖지 않는 단일 유전자로서 cas9를 검출했다. 이러한 3개의 유전자 좌의 경우, 본 발명자들은 cas9 유전자의 업스트림 또는 다운스트림의 임의의 작은 RNA 서열을 예상하지 못했다. R. 알부스 및 P. 엑크리멘티호미니스의 경우에, cas9를 함유하는 게놈 콘티그는 너무 짧아서 반복체 스페이서 어레이의 예상을 허용할 수 없다.
깊은 RNA 서열분석은 신규 tracrRNA 오쏘로그의 발현을 확인한다
인실리코 tracrRNA 예측을 증명하고, tracrRNA:전-crRNA 공처리 패턴을 결정하기 위해, 그람-양성(에스. 뮤탄스 및 엘. 이노쿠아) 및 그람-음성(엔. 메닌자이티디스, 씨. 제주니 및 에프. 노비시다) 박테리아로부터 선택된 RNA를 깊은 서열분석으로 분석하였다. tracrRNA 오쏘로그의 서열 및 처리된 crRNA를 회수했다(도 36 및 도 37). 에스. 파이오제네스26에서 이전에 공개된 차별적인 tracrRNA 서열분석 데이타와 일치하여, tracrRNA 오쏘로그는 총 맵핑된 판독치의 0.08 내지 6.2% 범위의 라이브러리로 크기 제시되었다. 처리된 tracrRNA는 또한 tracrRNA 판독치의 총량의 66% 내지 95% 이상의 범위로, 1차 전사체보다 더욱 풍부하였다(도 36 및 도 37).
도 36은 깊은 RNA 서열분석으로 나타낸 박테리아 tracrRNA 오쏘로그 및 crRNA의 발현을 묘사한다. 선택된 박테리아 균주의 tracrRNA 오쏘로그 및 crRNA의 발현 프로파일은 막대 차트로 상응하는 게놈(통합 유전체학 뷰어(IGV) 도구로부터 포획된 이미지)에 따라 제시된다. 캠필로박터 제주니 (GenBank: NC_002163), 프란시셀라 노비시다 (GenBank: NC_008601), 나이세리아 메닌자이티디스 (GenBank: NC_003116), 리스테리아 이노쿠아 (GenBank: NC_003212) 및 스트렙토코쿠스 뮤탄스 (GenBank: NC_004350). 게놈 배위체가 제시된다. a서열 적용범위는 BEDTools-버전-2.15.0(백만당 판독치로 제공된 규모)을 사용하여 계산했다. b각각의 뉴클레오타이드 위치에서 개시(5') 및 종결(3')하는 판독치의 분포도가 명시된다(판독치의 수로 제공된 규모). 상부 패널은 양성 가닥으로부터의 전사체에 상응하고, 하부 패널은 음성 가닥으로부터의 전사체에 상응한다. 축 아래에 제공된 음성 적용범위 값 및 피크는 게놈의 음성 가닥으로부터 전사를 나타낸다. 판독치의 두드러진 5'- 및 3'-말단이 모든 RNA에 대해 플롯팅된다. 엘. 이노쿠아 cDNA 라이브러리의 저품질이 제공되고, 판독치는 crRNA에 대해 단축되고, tracrRNA의 3' 말단에서 판독치의 축적이 아마도 RNA 분해에 기인하여 관찰된다는 것을 주시한다.
tracrRNA 1차 전사체의 5' 말단을 평가하기 위해, 본 발명자들은 tracrRNA의 모든 5' 말단 판독치의 풍부함을 분석하였고, 예상된 항-반복체 서열의 5' 말단 업스트림 또는 부근에서 가장 두드러진 판독치를 회수했다. tracrRNA 오쏘로그의 5' 말단을 프로모터 예측 알고리즘을 사용하여 추가로 확인하였다. 에스. 뮤탄스, 엘. 이노쿠아 및 엔. 메닌자이티디스로부터 확인된 tracrRNA의 5' 말단은 tracrRNA 발현의 인실리코 예측 및 노던 블랏 분석 둘 다와 서로 관련된다26. 씨. 제주니 tracrRNA의 가장 두드러진 5' 말단은 항-반복체 서열의 중간에서 확인되었다. 인실리코 예측과 서로 연관되고 CRISPR 반복 서열과의 상호작용하는 보다 긴 서열을 제공하는 5개의 뉴클레오타이드 업스트림 추가의 추정 5' 말단을 검출시켰다. 본 발명자들은 거의 배타적으로 처리된 전사체에 상응하는 에프. 노비시다 라이브러리로부터 비교적 적은 양의 판독치를 회수했다. 제 1 전사체의 매우 소량의 판독치의 분석은 강한 인실리코 프로모터 예측에 상응하는 5' 말단을 제공했다. 에프. 노비시다 tracrRNA의 노던 블랏 탐침검사는 길이 약 90nt의 전사체의 낮은 풍부함을 나타내는 예측의 정당성을 추가로 확인했다. 결과는 표 2에 열거된다. 모든 시험된 종에 대해, 엔. 메닌자이티디스를 제외하고, 1차 tracrRNA 전사체는 길이 75 내지 100nt의 단일 작은 RNA 종으로서 확인되었다. 엔. 메닌자이티디스의 경우, 본 발명자들은 약 110nt의 두드러진 1차 tracrRNA 형태 및 매우 낮은 양의 판독치로 제시되고 노던 블랏 분석에 의해 약한 밴드로서 이전에 검출된 약 170nt의 추정성 보다 긴 전사체를 발견했다.
[표 2]
선택된 tracrRNA 오쏘로그
tracrRNA 및 전-crRNA 공동-처리 부위는 항-반복체:반복체 영역에 존재한다.
본 발명자들은 예측된 항-반복체 서열 및 풍부한 성숙 crRNA 3’말단(도 34 및 45) 내에서 풍부한 tracrRNA 5’말단을 분석하여 처리된 tracrRNA 전사체를 시험했다. 모든 종에서, 본 발명자들은 RNase III에 의한 tracrRNA:전-crRNA 반복체 듀플렉스의 공동-처리로부터 생성될 수 있는 tracrRNA 오쏘로그의 두드러진 5' 말단을 확인했다. 본 발명자들은 또한 이전 관찰과 일관되게, 대부분 아마 추정 트리밍에 의한 제 2 성숙 이벤트로부터 생성되는 crRNA의 처리된 5'-말단을 확인했다. 주목할만한, 에스. 파이오제네스, 에스. 뮤탄스 및 엘. 이노쿠아의 밀접하게 관련된 RNA 쌍에서, 본 발명자들은 항-반복체 서열의 중간에서 G:C 염기쌍 주위의 동일한 처리 부위를 관찰했다. 에스. 뮤탄스 및 엘. 이노쿠아 둘 다에서, 본 발명자들은 tracrRNA:crRNA 듀플렉스의 추가의 트리밍을 제시할 수 있는 추가의 두드러진 tracrRNA 5’말단 및 crRNA 3' 말단을 검출하였고, crRNA의 3'-말단은 RNase III-촉매된 제 1 처리 이벤트 후, 이미 언급된 5'-말단 트리밍에서 추가로 단축된다. 유사하게, 씨. 제주니에서, 본 발명자들은 RNase III 처리 패턴에 적합화된 단지 소량의 crRNA 3’말단을 발견하였고, 처리된 tracrRNA의 상응하는 5' 말단을 회수했다. 따라서, RNase III에 의한 초기 절단 후 tracrRNA:crRNA 듀플렉스의 추정 트리밍은 성숙한 crRNA에서 보다 짧은 반복체 유도된 파트를 생성하여 엔도뉴클레아제 Cas9와 표적 DNA의 후속적인 절단의 상호작용을 위한 삼중 G:C 염기-짝짓기에 의해 안정화된 보다 짧은 tracrRNA:crRNA 듀플렉스를 생성한다. 엔. 메닌자이티디스 RNA 듀플렉스는 CRISPR 반복체의 3' 말단에서 더 멀리 두 개의 1차 부위에서 처리되어 듀플렉스 내의 중앙 중가에도 불구하고 성숙한 crRNA 중의 긴 반복체 유도된 파트 및 안정한 RNA:RNA 상호작용을 유도하는 것으로 나타난다. 흥미롭게도, 에프. 노비시다의 tracrRNA:전-crRNA 듀플렉스는 낮은 상보성 영역 내에서 절단되는 것으로 나타나고, tracrRNA의 회수된 풍부한 5' 말단의 일부는 crRNA의 공존 트리밍 없이 이의 추가의 트리밍을 제안한다. 1차 전사체 크기 및 처리 부위의 위치의 차이는 약 65 내지 85nt의 다양한 길이의 처리된 tracrRNA를 유도한다. 두드러진 처리된 tracrRNA 전사체의 좌표 및 크기는 표 2 및 도 37에 제시된다. tracrRNA 및 crRNA의 관찰된 처리 패턴은 이전에 제안된 두 성숙 이벤트 모델과 충분히 일치한다. tracrRNA 5'-말단 및 crRNA 3'-말단의 일부의 추정 추가 트리밍은 제 2 성숙 이벤트로부터 유래할 수 있거나, 대안적으로 cDNA 라이브러리 제조 또는 RNA 서열분석의 인공물일 수 있다. 이러한 처리의 특성은 추가로 조사된다.
tracrRNA 오쏘로그의 서열은 매우 다양하다
선택된 tracrRNA 오쏘로그의 서열 유사성이 또한 결정되었다. 본 발명자들은 에스. 파이오제네스 (89nt 형태 단독), 에스. 뮤탄스, 엘. 이노쿠아 및 엔. 메닌자이티디스 (110nt 형태 단독), 에스. 서모필러스, 피. 멀토시다 및 엠. 모빌레의 1차 tracrRNA 전사체의 다중 서열 정렬을 수행하였다(표 2, 도 35). 본 발명자들은 tracrRNA 서열의 높은 다양성을, 그러나 밀접하게 관련된 CRISPR-Cas 유전자 좌로부터 서열의 중요한 보존을 관찰했다. 엘. 이노쿠아, 에스. 파이오제네스, 에스. 뮤탄스 및 S. 써모필레스로부터의 tracrRNA는 평균 77% 동일성을 공유하고, 엔. 메닌자이티디스 및 피. 멀토시다로부터의 tracrRNA는 쌍 정렬에 따라 82% 동일성을 공유한다. 분석된 tracrRNA 서열의 평균 동일성은 랜덤 RNA 서열의 동일성과 비교할만한 56%이다. 이 관찰은 추가로 서열 유사성을 기반으로 하는 tracrRNA 오쏘로그의 예측이 밀접하게 관련된 유전자 좌의 경우에서만 수행될 수 있음을 확인한다. 본 발명자들은 또한 가능한 tracrRNA 구조 보존을 추구하였지만, 하나의 공동-변화 및 보존된 전사 종결자 구조 이외에는 임의의 중요한 유사성을 찾지 못했다(도 35).
도 35는 tracrRNA 오쏘로그의 서열 다양성을 묘사한다. tracrRNA 서열 다중 정렬. 에스. 서모필러스 및 에스. 서모필러스2, 따라서 서열번호 41 및 서열번호 40 Cas9 오쏘로그와 연관된 tracrRNA 블랙, 고도로 보존됨; 짙은 회색, 보존됨; 밝은 회색, 약하게 보존됨. 예상된 공유 서열 구조가 정렬의 상단부에 묘사된다. 화살표는 뉴클레오타이드 공동변화를 나타낸다. 에스. 파이오제네스 SF370, 에스. 뮤탄스 UA159, 엘. 이노쿠아 Clip11262, 씨. 제주니 NCTC 11168, 에프. 노비시다 U112 및 엔. 메닌자이티디스 A Z2491 tracrRNA는 RNA 서열분석 및 노던 블랏 분석으로 확인되었다. S. 써모필레스 LMD-9 tracrRNA는 노던 블랏 분석으로 확인되었다. 피. 멀토시다 Pm70 tracrRNA는 엔. 메닌자이티디스 A Z2491의 것과 함께 CRISPR-Cas 유전자 좌의 높은 유사성으로부터 예측했다. 엠. 모빌레 163K tracrRNA는 전사 프로모터 및 종결자의 강한 예측으로부터 인실리코 예측되었다.
실시예 4: 유전자 발현의 서열 특이적 조절을 위한 RNA 유도된 플랫폼으로서 CRISPR 용도 변경
게놈 전체에 대한 표적화된 유전자 조절은 세포성 시스템을 질문하고, 교란하고 조작하기 위한 강력한 전략이다. 본 발명자들은 유형 II CRISPR 시스템으로부터 Cas9, RNA-유도된 DNA 엔도뉴클레아제를 기본으로 하는 유전자 발현을 조절하는 신규 방법을 개발하였다. 이 실시예는 엔도뉴클레아제 활성이 부족한 촉매적으로 죽은 Cas9는, 가이드 RNA로 공발현될 경우, 전사 신도, RNA 폴리머라제 결합 또는 전사 인자 결합을 구체적으로 간섭할 수 있는 DNA 인식 복합체를 생성한다는 것을 입증한다. CRISPR 간섭(CRISPRi)이라 칭명되는 이 시스템은 검출가능한 부정확한 효과를 갖지 않는 에스케리치아 콜리에서 표적화된 유전자의 발현을 효율적으로 억제할 수 있다. CRISPRi를 사용하여 다중 표적 유전자를 동시에 억제할 수 있고, 이의 효과는 가역적이다. 또한, 상기 시스템은 포유동물 세포에서 유전자 억제에 적응될 수 있다. 이 RNA-유도된 DNA 인식 플랫폼은 게놈 전체 규모에 대해 유전자 발현을 선택적으로 교란시키는 단순한 접근법을 제공한다.
물질 및 방법
균주 및 배지
에스케리치아 콜리 K-12 균주 MG1655는 생체내 형광 측정을 위한 숙주 균주로서 사용되었다. RpoC 서브유닛의 C-말단 말단에 부착된 3x-FLAG 에피토프 태그로 RNAP의 변이체를 내생적으로 발현시키는 이. 콜라이 MG1655-유도된 균주를 모든 서열분석 실험에 사용했다. EZ 풍부한 규정된 배지(EZ-RDM, Teknoka)를 생체내 형광 분석용 성장 배지로서 사용했다. 유전적 형질전환 및 형질전환의 입증은 표준 프로토콜을 사용하고 선택가능한 마커로서 AmpR, CmR, 또는 KanR 유전자를 사용하여 수행하였다.
플라스미드 구성 및 이. 콜라이 게놈 클로닝
Cas9 및 dCas9 유전자를 각각 이전에 기재된 벡터 pMJ806 및 pMJ841로부터 클로닝하였다. 유전자를 PCR 증폭시키고, 언하이드로테트라사이클린(aTc)-유도성 프로모터 PLtetO-1, 클로르암페니콜 선택가능한 마커 및 p15A 복제 개시점을 함유하는 벡터에 삽입했다. sgRNA 템플레이트를 주석이 달린 전사 개시 부위, 암피실린 선택가능한 마커 및 ColE1 복제 개시점과 함께 최소 합성 프로모터(J23119)를 함유하는 벡터에서 클로닝했다. 역 PCR을 사용하여 새로운 20-bp 상보적 영역을 갖는 sgRNA 카세트를 생성하였다. 형광 리포터 유전자를 이. 콜라이에 삽입하기 위해, 형광 유전자를 먼저 진입 벡터 위에서 클로닝하고, 이어서 이를 PCR 증폭시켜 nsfA 5'/3' UTR 서열, 형광 유전자 및 KanR 선택가능한 마커를 함유하는 선형화된 DNA 단편을 생성했다. 이. 콜라이 MG1655 균주를 λ-Red 재조합 단백질(엑소, 베타 및 감마)을 함유한 온도-민감성 플라스미드 pKD46로 형질전환시켰다. 세포 배양물을 30℃에서 OD(600nm) 약 0.5로 성장시키고, 0.2% 아라비노오스를 부가하여 1시간 동안 λ-Red 재조합 단백질의 발현을 유도했다. 세포를 4℃에서 수확하고, 전기천공에 의한 선형화 DNA 단편의 형질전환용으로 사용했다. 정확한 게놈 삽입을 함유하는 세포를 50㎍/mL 카나마이신을 사용하여 선택했다.
유세포측정 및 분석
균주를 37℃ 및 1200rpm에서 밤새 2mL 96-웰 깊은 웰 플레이트(Costar 3960)에서 100㎍/mL 카르베니실린 및 34㎍/mL 클로르암페니콜을 함유하는 EZ-RDM에서 배양했다. 이어서, 이 밤새 배양액 1μL를 dCas9 단백질의 생산을 유도하기 위해 보충된 2μM aTc와 함께 동일한 항생제 농도로 249μL의 신선한 EZ-RDM에 부가하였다. 세포가 중간-대수 단계로 성장시켰을 때(약 4h), 형광 단백질의 수준은 고-처리량 샘플러가 장착된 LSRII 유세포 계측기(BD Biosciences)를 사용하여 측정하였다. 세포를 적어도 20,000세포가 수집될 때까지 낮은 유속으로 샘플링하였다. 데이타를 자방 산란기 측면 산란기 플롯에 60% 세포 집단을 함유하는 다각형 영역 상에서 게이팅하여 FCS Express(드노보 소프트웨어)를 사용하여 분석했다. 각 실험을 위해, 삼중 배양물을 측정하고, 그들의 표준 편차를 오차 막대로서 명시했다.
B-갈락토시다아제 검정
β-갈락토시다아제 검정을 수행하기 위해, 상기와 같이 제조된 1μL의 밤새 배양물을 1mM 이소프로필 β-D-1-티오갈락토피라노사이드(IPTG)를 포함하거나 포함하지 않고 2μM aTc와 함께 동일한 항생제 농도로 249μL의 신선한 EZ-RDM에 부가하였다. 세포를 중간-대수 단계로 성장시켰다. 이 배양물 100μL의 LacZ 활성을 지침에 따라 효모 β-갈락토시다아제 검정 키트(Pierce)를 사용하여 측정하였다.
총 RNA의 추출 및 정제
각 샘플을 위해, 이. 콜라이의 모노클로날 배양물을 37℃에서 500mL의 EZ-RDM 중의 OD(600nm) 0.1로부터 초기 대수 단계(OD 0.45 ± 0.05)로 성장시키고, 이 시점에 세포를 0.22㎛ 니트로셀룰로오스 필터(GE)를 통해 여과로 수확하고, 액체 질소로 냉동시켜 모든 전사 과정을 동시에 중단시켰다. 냉동된 세포(100㎍)를 10 mM MnCl2 및 15μM 타게틴 전사 억제제(Epicentre)가 보충된 500μL의 냉동된 용해 완충제(20 mM 트리스 pH 8, 0.4% 트리톤 X-100, 0.1% NP-40, 100 mM NH4Cl, 50 U/mL SUPERase·In (Ambion) 및 1x 프로테아제 억제제 칵테일(완전함, EDTA-부재, Roche)의 존재하에 Qiagen TissueLyser II 혼합기 상에서 15Hz에서 3분 동안 6회 분쇄했다.
용해물을 피펫팅하여 얼음 위에 재현탁시켰다. RQ1 DNase I(110 U 총, Promega)를 부가하고, 얼음 위에서 20분 동안 배양했다. 반응물을 EDTA(25mM 최종)로 급냉시키고, 용해물을 20,000g에서 10분 동안 원심분리시켜 4℃에서 정화시켰다. 용해물을 PD MiniTrap G-25 칼럼(GE Healthcare) 상에 부하하고, 1mM EDTA로 보충된 용해 완충제로 용출시켰다.
총 mRNA 정제
총 RNA는 miRNeasy 키트(Qiagen)를 사용하여 정화된 용해물로부터 정제시켰다. 20μL의 10mM 트리스 pH 7 중의 1㎍의 RNA를 동일 용적의 2x 알칼리성 단편화 용액(2mM EDTA, 10mM Na2CO3, 90mM NaHCO3, pH 9.3)과 혼합하고, 95℃에서 약 25분 동안 배양하여 30 내지 100nt 범위의 단편을 생성하였다. 단편화 반응은 0.56mL의 빙냉 침전 용액(300mM NaOAc pH 5.5 + 글리코블루(Ambion))을 첨가하여 정지시키고, RNA는 표준 이소프로판올 침전으로 정제시켰다. 이어서, 단편화된 mRNA를 50μL의 반응물 중에서 1x PNK 완충제(ATP 없음) + 0.5U SUPERase·In 중의 25 U T4 PNK(NEB)로 탈포스포릴화하고, 표준 이소프로판올 침전 방법을 통해 글리코블루로 침전시켰다.
초기 RNA 정제
초기 RNA 정제를 위해, 정화된 용해물을 이전에 기재된 바와 같이 0.5mL 항-FLAG M2 친화성 겔(시그마 알드리치)에 부가하였다. 친화성 겔을 장동과 함께 4℃에서 2.5시간 동안 정화된 용해물로 배양 전에 1mM EDTA로 보충된 용해 완충제로 2회 세척하였다. 면역침전을 300mM KCl이 보충된 용해 완충제로 4 x 10ml 세척하고, 결합된 RNAP를 1 mM EDTA 및 2 mg/mL 3x-FLAG 펩타이드 (시그마 알드리치)가 보충된 용해 완충제로 2회 용출시켰다. 초기 RNA를 miRNeasy 키트(Qiagen)를 사용하여 용출액으로부터 정제하고, 이전에 확립된 라이브러리 발생 프로토콜을 DNA로 전환시켰다.
DNA 라이브러리 제조 및 DNA 서열분석
DNA 라이브러리를 일루미나 HiSeq 2000 상에서 서열분석했다. 판독치를 HTSeq 파이썬 패키지 및 파이썬으로 작성된 기타 사용자 소프트웨어를 사용하여 처리했다. 서열분석된 전사체의 3'-말단을 보우타이("보우타이-바이오" 처리 ".sourceforge.net") 및 MochiView로 생성된 RNAP 프로파일("johnsonlab.ucsf" 처리 ".edu/mochi.html")을 사용하여 참조 게놈에 정렬시켰다.
인간 세포에서 CRISPRi를 위한 플라스미드 설계 및 구성
포유동물 코돈 최적화된 스트렙토코쿠스 파이오제네스 Cas9(DNA 2.0)를 인코딩하는 서열을 3개의 C-말단 SV40 핵 국재화 서열(NLS)을 사용하여 또는 두 개의 NLS로 플랭킹된 tagBFP에 융합시켰다. 표준 결찰 독립적 클로닝을 사용하여, 본 발명자들은 이러한 두 융합 단백질을 MSCV-Puro(Clontech)에 클로닝했다. 가이드 sgRNA를 CMV 프로모터로부터 mCherry를 공-발현시키는 pSico로부터 유도된 렌티바이러스 U6 기반 발현 벡터를 사용하여 발현시켰다. sgRNA 발현 플라스미드를 어닐링된 프라이머를 BstXI 및 XhoI로 소화된 렌티바이러스 U6 기반 발현 벡터로 삽입하여 클로닝하였다.
인간 세포 중의 CRISPRi를 위한 세포 배양, DNA 형질감염 및 형광 측정
HEK293 세포를 10%FBS, 2mM 글루타민, 100units/mL 스트렙토마이신 및 100㎍/mL 페니실린 중의 둘베코 변형 이글 배지 (DMEM)에서 유지시켰다. HEK293을 표준 프로토콜을 사용하여 MSCV 레트로바이러스를 발현시키는 GFP로 감염시키고, 안정한 GFP 발현을 위해 BD FACS Aria2를 사용하여 유세포 계측법으로 분류했다. HEK293 세포를 발현시키는 GFP를 24웰 플레이트에서 0.5㎍의 dCas9 발현 플라스미드 및 0.5㎍의 RNA 발현 플라스미드(도 45B에 대해 0.25㎍의 GFP 리포터 플라스미드 포함)를 사용하여 제조업자 권장된 프로토콜과 함께 TransIT-LT1 형질감염 시약(Mirus)을 사용하여 일시적으로 형질감염시켰다. 형질감염 후 72시간에, 세포를 단일 세포 현탁액으로 트립신화하였다. U6 벡터는 mCherry 유전자를 구동하는 구성적 CMV 프로모터를 함유한다. GFP 발현을 mCherry 양성 모집단(음성 대조군 세포에 대해 >10배 더 밝은 mCherry) 상에서 게이팅하여 BD LSRII FACS 기계를 사용하여 분석했다.
설계된 RNA
도면에 사용된 sgRNA 설계: 단지 20개 뉴클레오타이드 일치 영역(DNA 표적화 세그먼트)이 열거된다(달리 주시되지 않으면):
도 40c에 사용된 mRFP-표적화 sgRNA(서열번호 741-746);
도 40d에 사용된 프로모터-표적화 sgRNA(서열번호 747-751);
도 40d 중의 표적 프로모터 서열(서열번호 752);
도 43b에 사용된 mRFP-표적화 sgRNA(서열번호 753-760);
도 42b에 사용된 sfGFP-표적화 sgRNA (gfp)(서열번호 761);
도 43b에 사용된 sfGFP-표적화 sgRNA(서열번호 762-769);
도 43f 및 도 51 중의 이중-sgRNA 표적화 실험(서열번호 770-778);
도 44b에 사용된 lac 오페론-표적화 sgRNA(서열번호 779-787); 및
도 45에 사용된 EGFP-표적화 sgRNA(서열번호 788-794).
[표 3]
실시예 4(상기 열거됨)의 도면에 사용된 서열
결과
CRISPR(짧은 회귀성 반복체 사이에 규칙적으로 공간을 두고 클러스터된) 시스템은 표적화된 유전자 조절을 위한 새로운 잠재적인 플랫폼을 제공한다. 약 40%의 박테리아 및 90%의 고세균은 외래 DNA 요소에 내성을 부여하기 위해 CRISPR/CRISPR-연관된(Cas) 시스템을 포함한다. CRISPR 시스템은 서열 특이적 방식으로 외래 DNA 요소를 표적화하고 절단하기 위해 작은 염기 짝짓기 RNA를 사용한다. 상이한 유기체에 다양한 CRISPR 시스템이 존재하고, 가장 간단한 것 중의 하나는 스트렙토코쿠스 파이오제네스로부터 유형 II CRISPR 시스템이고: Cas9 단백질 및 두 개의 RNA를 인코딩하는 유일한 단일 유전자, 성숙한 CRISPR RNA(crRNA) 및 부분적으로 상보적인 트랜스-작용상 RNA(tracrRNA)가 외래 DNA의 RNA-유도된 사이런싱에 필요하고 충분하다(도 46). crRNA의 성숙은 tracrRNA 및 RNase III를 필요로 한다. 그러나, 이 요건은 tracrRNA-crRNA 복합체를 모방하는 설계된 헤어핀을 함유하는 조작된 작은 가이드 RNA(sgRNA)를 사용하여 우회할 수 있다. sgRNA와 표적 DNA 사이의 염기 짝짓기는 Cas9의 엔도뉴클레아제 활성에 기인하여 이중-가닥 파괴(DSB)을 유발한다. 결합 특이성은 sgRNA-DNA 염기 짝짓기 및 DNA 상보적 영역과 병렬된 짧은 DNA 모티프(프로토스페이서 인접 모티프 또는 PAM, 서열: NGG) 둘 다에 의해 결정된다. 따라서, CRISPR 시스템은 단지 두 분자-Cas9 단백질 및 sgRNA의 최소 세트를 필요로 하고, 따라서 전위를 숙주 독립적 유전자 표적화 플랫폼으로서 사용되도록 유지한다. Cas9/CRISPR은 부위-선택적 RNA-유도된 게놈 편집 (도 39a)에 이용할 수 있다는 것이 증명되었다.
도 46은 에스. 파이오제네스로부터 유형 II CRISPR 시스템의 기전을 묘사한다. 상기 시스템은 CRISPR-연관된(Cas) 단백질 및 반복체 스페이서 서열의 어레이를 함유하는 CRISPR 유전자 좌의 세트로 이루어진다. 모든 반복체는 동일하고, 모든 스페이서는 상이하고 표적 DNA 서열에 상보적이다. 세포가 외래 DNA 요소로 감염될 경우, CRISPR 유전자 좌는 긴 전구체 전사체로 전사하고, 이는 보다 작은 단편으로 절단된다. 절단은 처리 안티센스 RNA(tracrRNA) 및 숙주 RNase III에 의해 매개된다. 절단 후, 하나의 단일 단백질, Cas9를 인식하고, crRNA의 절단된 형태에 결합한다. Cas9는 crRNA를 DNA에 가이드하고, DNA 분자를 스캔한다. 복합체는 crRNA과 DNA 표적 사이의 염기 짝짓기에 의해 안정화된다. 이 경우에, Cas9는 이의 뉴클레아제 활성에 기인하여 이중-가닥 DNA 파괴를 유발한다. 이는 일반적으로 동족 DNA 분자를 제거하고, 세포는 특정 DNA 모집단에 면역성을 부여한다.
도 39는 CRISPR 간섭(CRISPRi) 시스템의 설계를 묘사한다. (A) 최소 간섭 시스템은 단일 단백질 및 설계된 sgRNA 키메라로 이루어진다. sgRNA 키메라는 3개의 도메인(박스 영역): 특이적 DNA 결합을 위한 20-뉴클레오타이드 (nt) 상보적 영역, Cas9 결합(Cas9 핸들)을 위한 42-nt 헤어핀 및 에스. 파이오제네스로부터 유도된 40-nt 전사 종결자로 이루어진다. 야생형 Cas9 단백질은 뉴클레아제 활성을 함유한다. dCas9 단백질은 뉴클레아제 활성이 부족하다. (B) 야생형 Cas9 단백질은 sgRNA에 결합하고, 단백질-RNA 복합체를 형성한다. 복합체를 sgRNA와 DNA 표적 사이의 왓슨-크릭(Watson-Crick) 염기 짝짓기로 특이적 DNA 표적에 결합한다. 야생형 Cas9의 경우에, DNA는 Cas9 단백질의 뉴클레아제 활성에 기인하여 절단된다. 뉴클레아제 부족한 Cas9의 경우에, 복합체는 적합한 전사를 분열시킨다.
최소 CRISPRi 시스템은 단일 단백질 및 RNA로 이루어지고, 전사 개시 및 스트레치를 효과적으로 침묵시킬 수 있다
이. 콜라이 중의 이러한 CRISPRi 플랫폼을 실행하기 위해, 야생형 에스. 파이오제네스 Cas9 유전자 및 sgRNA를 시스템이 표적화 유전자 좌(도 40a)에서 유전자 발현을 교란할 수 있는지를 결정하기 위해 박테리아 벡터로부터 발현시켰다. 에스. 파이오제네스 CRISPR 시스템은 천연 이. 콜라이 시스템에 직각이다. Cas9 단백질은 p15A 복제 개시점을 함유하는 플라스미드 상에서 언하이드로테트라사이클린(aTc)-유도성 프로모터로부터 발현되고, sgRNA는 ColE1 복제 개시점을 함유하는 플라스미드 상에서 최소 구성적 프로모터로부터 발현된다. 대안적인 전략으로서, DNA 절단에서 부족한 촉매적으로 죽은 Cas9 돌연변이체(dCas9)가 사용되었고, Cas9의 이 형태가 단순한 RNA-유도된 DNA 결합 복합체로서 작용한다는 것을 보여주었다.
도 40은 CRISPRi가 전사 스트레치 및 개시를 효과적으로 침묵시킨다는 것을 입증한다. (A) CRISPRi 시스템은 유도성 Cas9 단백질 및 설계된 sgRNA 키메라로 이루어진다. dCas9는 RuvC1 및 HNH 뉴클레아제 도메인의 돌연변이를 함유한다. sgRNA 키메라는 도 1에 기재된 바와 같은 3개의 기능성 도메인을 함유한다. (B) 설계된 sgRNA(NT1) 및 DNA 표적의 서열. NT1은 mRFP 코딩 영역의 비-템플레이트 DNA 가닥을 표적화한다. 염기 짝짓기 모티프(20-nt)를 둘러싸는 영역만이 제시된다. 염기 짝짓기 뉴클레오타이드에 번호를 붙이고, dCas9-결합 헤어핀은 윗줄친다. PAM 서열은 밑줄친다. (C) CRISPRi는 전사 스트레치를 가닥-특이적 방식으로 차단했다. mRFP-코딩 유전자를 함유하는 합성 형광-기반 리포터 시스템을 이. 콜라이 MG1655 게놈(nsfA 유전자 좌)에 삽입하였다. 템플레이트 DNA 가닥 또는 비-템플레이트 DNA 가닥에 결합하는 6개의 sgRNA를 dCas9 단백질로 공발현시켰고, 표적 mRFP에 대한 그들의 효과를 생체내 형광 검정으로 측정했다. 비-템플레이트 DNA 가닥에 결합하는 유일한 sgRNA는 사일런싱(10 내지 300배)을 나타냈다. 대조군은 dCas9 단백질을 갖지만 sgRNA는 갖지 않는 세포의 형광성을 나타낸다. (D) CRISPRi는 전사 개시를 차단한다. 5개의 sgRNA가 이. 콜라이 프로모터(J23119) 주위의 상이한 영역에 결합하도록 설계되었다. 전사 개시 부위는 +1로서 표지되었다. 점선 타원은 -55 내지 +20의 75-bp 영역을 포함하는 초기 RNAP 복합체를 나타낸다. 초기 RNAP 복합체 내부의 유일한 sgRNA 표적화 영역은 억제(P1-P4)를 나타냈다. 전사 스트레치 블록과 달리, 사일런싱은 표적화된 DNA 가닥과 독립적이다. (E) CRISPRi 조절은 가역적이었다. dCas9 및 sgRNA(NT1) 둘 다는 aTc-유도성 프로모터의 조절하에 존재했다. 세포 배양물을 지수 단계 동안 유지시켰다. 시간 T = 0에서, 1μM의 aTc를 OD = 0.001인 세포에 보충했다. 표적 mRFP의 억제는 10분 이내에 개시했다. 형광 신호는 세포 성장과 일치하는 방식으로 감소되고, 상기 감소는 세포 분열에 기인한다는 것을 시사한다. 240분에, 형광성은 완전히 억제된 수준에 도달했다. T = 370분에, aTc는 성장 배지로 세척하고, 세포를 OD = 0.001로 역 희석시켰다. 형광성은 50분 후에 증가하기 시작하였고, 양성 대조군과 동일 수준으로 상승시키는데 약 300분이 소요되었다. 양성 대조군: 항상 유발제 없음; 음성 대조군: 항상 1μM aTc 유발제 포함. 2C, 2D 및 2E에서 형광성 결과는 적어도 3개의 생물학적 복제물의 평균 및 SEM을 나타낸다. 또한 도 47 및 도 48 참조.
Cas9와 공발현된 sgRNA 분자는 3개의 세그먼트: 20-뉴클레오타이드 (nt) 표적 특이적 상보적 영역, 42-nt Cas9 결합 헤어핀(Cas9 핸들) 및 에스. 파이오제네스로부터 유도된 40-nt 전사 종결자(도 40b)로 이루어진다. 레드 형광 단백질(mRFP)-기반 리포터 시스템을 이. 콜라이 MG1655 게놈에 삽입했다.
mRFP 코딩 서열에 표적화된 야생형 Cas9 단백질 및 sgRNA (NT1)의 공발현은 아마 게놈 상의 Cas9 유도된 이중-가닥 파괴 때문에 형질전환 효율을 극적으로 감소시켰다(도 47a). 약간의 생존자 콜로니의 서열분석은 그들이 모두 게놈 상의 표적 mRFP 부위 주위에 서열 재배열을 갖고 숙주 세포에 표적화된 야생형 Cas9 및 sgRNA의 발현에 대한 강한 선택이 존재했음을 시사한다는 것을 나타냈다. RuvC1 및 HNH 뉴클레아제 도메인(D10A 및 H841A)의 두 사일런싱 돌연변이를 함유한 dCas9 돌연변이체 유전자(비-절단성)은 형질전환 효율 및 이. 콜라이 성장률(도 47a & b)에 의해 입증된 바와 같이 이 치사율을 경감시켰다.
도 47은 도 40과 관련되고, dCas9 및 sgRNA로 공-형질전환된 이. 콜라이 세포 배양물의 성장 곡선을 나타낸다. (A) 이. 콜라이 세포를 두 플라스미드로 형질전환하기 위한 형질전환 효율. 하나의 플라스미드는 mRFP의 게놈 카피에 표적화하는 sgRNA를 함유하고, 다른 플라스미드는 야생형 Cas9 또는 dCas9를 함유한다. 야생형 Cas9 및 sgRNA의 공-형질전환은 매우 독성이고, 이는 dCas9를 사용하여 경감시킬 수 있다. (B) sgRNA (NT1)는 mRFP의 코딩 서열을 표적화하도록 설계된다. dCas9 및 sgRNA의 공발현은 세포 성장률에 대해 거의 어떤 효과도 나타내지 않아 DNA와의 dCas9-sgRNA 상호작용이 DNA 폴리머라제가 아니라 RNA 폴리머라제 또는 세포 복제를 차단하기에 충분히 강하다는 것을 시사한다. 결과는 적어도 3개의 독립적인 실험의 평균 및 SEM을 나타낸다.
dCas9:sgRNA 복합체가 유전자 발현의 매우 효율적인 억제를 산출할 수 있는지를 시험하기 위해, mRFP 코딩 서열의 상이한 영역에 상보적인 sgRNA는 템플레이트 DNA 가닥 또는 비-템플레이트 DNA 가닥에 결합하도록 설계되었다. 결과는, 비-템플레이트 DAN 가닥을 표적화하는 sgRNA가 효과적인 유전자 사일런싱(10 내지 300배의 억제)을 입증하였고, 반면 템플레이트 가닥을 표적화하는 것들은 약간의 효과를 나타냈다(도 40c). 상기 시스템은 이. 콜라이 게놈 이내에 고-카피 플라스미드 위에 존재한 유전자에 대해 유사한 억제 효과를 나타냈다(도 48). 더욱이, 프로모터 영역에서의 표적화는 또한 효과적인 유전자 사일런싱을 유도한다(도 40d). -35 박스에 대한 sgRNA의 표적화하는 유전자 발현을 상당히 녹다운시키고(P1, 약 100-배의 억제), 반면에 다른 인접한 영역에서의 표적화는 습윤 효과를 나타냈다(P2-P4). 프로모터의 약 100-bp 업스트림 서열의 표적화는 어떤 효과도 나타내지 않았다(P5). 코딩 서열의 표적화와 달리, 프로모터를 표적화할 때, 사일런싱의 효율은 DNA 가닥과 독립적이고, 템플레이트 또는 비-템플레이트 가닥의 표적화는 동등하게 효과적이다(P2 및 P3).
도 48은 도 40C에 관련되고, CRISPRi가 다중-카피 플라스미드 상에서 리포터 유전자의 발현을 침묵시킬 수 있음을 나타낸다. mRFP 유전자는 p15A 플라스미드 위에 클론화했다. dCas9 및 mRFP-특이적 sgRNA(NT1)의 존재는 mRFP를 강하게 억제한다(약 300-배). 억제 효과는 게놈에서 mRFP를 사용하여 관찰된 것과 유사하다(도 40c). 사일런싱은, sgRNA가 템플레이트 DNA 가닥(T1)이 아니라 비템플레이트 DNA 가닥에 작용할 때 단지 효과적이다. 또한, 사일런싱은, GFP-특이적 3 sgRNA(gfp)가 mRFP 발현에 어떤 효과도 나타내지 않기 ?문에, 매우 특이적이다. 형광성 결과는 적어도 3개의 생물학적 복제물의 평균 및 SEM을 나타낸다.
CRISPRi 유전자 녹다운은 유도성 및 가역적이다
유전자 녹아웃 방법과 달리, 유전자 발현의 CRISPRi-기반 녹 다운의 하나의 장점은 이 동요는 가역적이어야 한다는 사실이다. CRISPRi 조절이 유도되고 이어서 역전될 수 있는지를 시험하기 위해, dCas9 및 mRFP-특이적 sgRNA(NT1) 둘 다를 aTc-유도성 프로모터의 조절하에 배치하고, 유발제에 반응하여 mRFP의 CRISPRi-매개된 조절의 시간 경과 측정을 수행하였다(도 40e). 0시간에, 유발자 없이 조기 지수 단계에서 성장한 세포 배양물에 1μM의 aTc를 보충하였다. 상기 데이타는 유발제의 존재에 신속하게 반응할 수 있고-형광성 리포터 단백질 신호가 유발제 분자의 부가 10분 이내에 감소하기 시작했다는 것을 나타냈다. mRFP 단백질이 안정하기 때문에 형광성 신호 감소 속도는, 유사한 세포 배가 시간 및 형광성 반감기의 손실(둘 다 약 36분)로 알 수 있는 바와 같이, 세포 성장에 기인하는 단백질 희석에 의해 제한된다. 240분에, 모든 세포는 음성 대조군과 동일 수준으로 균일하게 억제되었다. 420분에, 유발제를 성장 배지로 세정하고, 세포를 낮은 OD로 역 희석시켰다. 50분 지연 후, mRFP 형광성은 증가하기 시작했다. 단일-세포 형광성을 양성 대조군과 동일 수준으로 증가시키는 데 총 300분이 소요되었다. 50분 지연은 대부분 아마 세포 성장 및 분열에 의한 희석으로 dCas9/sgRNA 턴오버 속도 오프셋에 의해 결정된다. 요약하면, 이러한 결과는 dCas9-sgRNA의 사일런싱 효과가 유도되고 역전될 수 있다는 것을 입증한다.
천연 스트레치 전사체 서열분석(NET-Seq)은 CRISPRi가 전사를 차단하여 기능한다는 것을 확인한다
dCas9는 전사 스트레치 동안 RNA 폴리머라제(RNAP)를 차단할 수 있는 RNA-유도된 DNA 결합 복합체로서 기능하는 것으로 나타났다. 비-템플레이트 DNA 가닥이 전사된 mRNA 및 비-템플레이트 DNA 가닥에 결합하는 단지 sgRNA가 사일런싱을 나타낼 때 동일한 서열 동일성을 공유하기 때문에, dCas9:sgRNA 복합체가 mRNA와 상호작용하고, 이의 번역 또는 안정성을 변경할 가능성이 잔류한다. 이러한 가능성을 구별하기 위해, 최근에 기재된 천연 스트레치 전사체 서열분석(NET-seq) 접근법을 이. 콜라이에 적용하였고, 이는 RNA 폴리머라제를 스트레치시키는 위치를 세계적으로 프로파일링하고 전사에 대한 dCas9:sgRNA 복합체의 효과를 모니터링하기 위해 사용될 수 있다. 이 NET-seq 방법에서, CRISPRi 시스템은 FLAG-태그된 RNAP를 함유한 이. 콜라이 MG1655-유도된 균주로 형질전환되었다. CRISPRi는 mRFP 코딩 영역에 결합하는 sgRNA(NT1)을 함유했다. RNAP의 스트레치와 관련되는 천연 전사체의 서열분석이 따르는 태그된 RNAP의 시험관내 면역정제는 RNAP의 정지 부위를 구별할 수 있게 했다.
이러한 실험은 sgRNA가 sgRNA 표적 유전자 좌(도 41a)의 강한 전사 중지 업스트림을 유도했다는 것을 입증한다. 중지 부위와 표적 부위 사이의 거리는 19-bp이고, 이는 RNAP의 뉴클레오타이드 통합과 그의 전단 사이에 이전에 기록된 약 18-bp 거리와 완벽하게 일치한다. 이 발견은 전사 블록이 스트레치 RNAP 및 dCas9:sgRNA 복합체(도 41b) 사이의 물리적 충돌에 기인하는 CRISPRi의 기전과 일치한다. 템플레이트 가닥에 dCas9:sgRNA 복합체의 결합은 약간의 억제 효과를 가져서 RNAP가 이 특별한 배향에서 복합체를 통해 판독할 수 있다는 것을 시사한다. 이러한 경우에, sgRNA는 RNAP에 직면하고, 이는 RNAP의 헬리카제 활성에 의해 압축을 풀 수 있다. 이러한 실험은, CRISPRi가 전사를 직접 차단하기 위해 RNA를 이용한다는 것을 입증했다. 이 기전은 유전자 발현의 녹다운이 이들의 번역 이전에 이미 전사된 메신저 RNA의 파괴를 필요로 하는 RNAi의 것과 구별된다.
도 41는 CRISPRi가 전사 스트레치를 차단하여 기능한다는 것을 입증한다. (A) FLAG-태그된 RNAP 분자를 면역침전시키고, 연관된 천연 mRNA 전사체를 서열분석했다. 최상부 패널은 sgRNA 없는 세포에서 천연 mRFP 전사체의 서열분석 결과를 나타내고, 하단부 패널은 sgRNA를 갖는 세포에서의 결과를 나타낸다. sgRNA의 존재하에, 강한 전사 중지는 표적 부위의 19-bp 업스트림을 관찰하였고, 이후 서열분석 판독치의 수는 급격하게 떨어진다. (B) 제안된 CRISPRi 기전은 RNAP와 dCas9-sgRNA 사이의 물리적 충돌을 기반으로 한다. RNAP의 중심으로부터 이의 전단까지의 거리는 약 19bp이고, 이는 전사 중지 위치와 sgRNA 염기 짝짓기 영역의 3' 사이의 본 측정된 거리와 잘 일치한다. 중지된 RNAP는 dCas9-sgRNA 장애물과 마주칠 때 전사 스트레치를 중단한다.
CRISPRi sgRNA-유도된 유전자 사일런싱은 매우 특이적이다
게놈 전체 규모에 대한 CRISPRi의 특이성을 평가하기 위해, sgRNA 공-발현을 갖거나 갖지 않는 dCas9-형질전환된 세포의 전체 전사체 샷건 서열분석(RNA-seq)을 수행하였다(도 42a). mRFP(NT1)에 표적화된 sgRNA의 존재하에, mRFP 전사체는 풍부함의 감소를 나타내는 유일한 유전자였다. 어떤 기타 유전자도 서열분석 오차내에서 sgRNA의 부가시 발현의 상당한 변화를 나타내지 않았다. 본 발명자들은 또한 상이한 유전자를 표적화하는 상이한 sgRNA를 갖는 세포 상에서 RNA-seq를 수행하였다. 이러한 실험 중의 어떤 것도 표적화된 유전자(도 49) 이외에 유전자의 상당한 변화를 나타내지 않았다. 따라서, sgRNA-유도된 유전자 표적화 및 조절은 매우 특이적이고, 중요한 부정확한 효과를 갖지 않는다.
도 42는 CRISPRi 시스템의 표적화 특이성을 입증한다. (A) 게놈-규모 mRNA 서열분석(RNA-seq)은 CRISPRi 표적화가 부정확한 효과를 갖지 않는다는 것을 확인했다. mRFP 코딩 영역에 결합하는 sgRNA NT1을 사용하였다. dCas9, mRFP 및 sfGFP 유전자가 강조된다. (B) 다중 sgRNA는 동일 세포에서 두 개의 형광성 단백질 리포터를 독립적으로 침묵시킬 수 있다. 각 sgRNA는 기타 유전자가 아니라 이의 동족 유전자를 특이적으로 억제했다. 두 sgRNA가 제공될 때, 두 유전자는 침묵되었다. 오차는 적어도 3개의 생물학적 복제물로부터 SEM을 나타낸다. (C) 두 개의 형광성 단백질을 조절하기 위해 두 개의 sgRNA를 사용하기 위한 현미경적 이미지. 최상부 패널은 이. 콜라이 세포의 명시야 이미지를 나타내고, 중간 패널은 RFP 채널을 나타내고, 하단부는 GFP 패널을 나타낸다. 하나의 sgRNA 및 dCas9의 공발현은 단지 기타가 아니라 동족 형광성 단백질을 침묵시킨다. 녹아웃 효과는 강했지만, 침묵된 특정 형광성 단백질을 갖는 세포로부터 거의 어떠한 형광성도 관찰되지 않았다. 규모 막대, 10㎛. 대조군은 임의의 형광성 단백질 리포터가 없는 세포를 나타낸다. 형광성 결과는 적어도 3개의 생물학적 복제물의 평균 및 SEM을 나타낸다. 또한 도 49를 참조한다.
도 49는 도 42a에 관련되고, 상이한 유전자를 표적화하는 sgRNA를 갖는 세포의 RNA-seq 데이타를 묘사한다. (A) 이. 콜라이에서 내인성 lacI 유전자의 프로모터를 표적화하는 (+/-) sgRNA. 동일한 lacI-표적화 sgRNA는 도 44a에서와 같이 사용되었다. (B) 자가-억제된 sgRNA가 없는 세포에 대한 (+/-) 1mM IPTG(sgRNA는 이의 자체 프로모터를 재억제한다). (C) 이. 콜라이에서 내인성 lacZ 유전자를 표적화하는 (+/-) sgRNA. 동일한 lacZ-표적화 sgRNA는 도 44a에서와 같이 사용되었다. 1mM IPTG가 또한 lacZ-표적화 sgRNA를 갖는 세포에 보충되었다.
CRISPRi는 다중 유전자를 동시에 조절하기 위해 사용될 수 있다
CRISPRi 시스템은 크로스토크 없이 독립적으로 다중 유전자의 조절을 가능하게 할 수 있다. mRFP 및 sfGFP를 기반으로 하는 이중 색상 형광-리포터 시스템이 설계되었다. 뚜렷한 상보적 영역을 갖는 두 개의 sgRNA가 설계되었다. 각 gRNA의 발현은 단지 동족 유전자를 침묵시키고, 기타에 대한 효과를 갖지 않았다. 두 sgRNA의 공발현은 두 유전자를 녹다운시켰다(도 42b & c). 이러한 결과는 sgRNA-유도된 표적화가 특이적임을 시사하고, 특이성은 이의 서열 동일성에 의해 지시되고, 기타 sgRNA의 존재로 영향받지 않는다. 이 거동은 동시에 CRISPRi에 의해 다중 유전자의 다중 제어를 가능하게 할 수 있어야 한다.
CRISPRi 사일런싱 효율을 측정하는 인자
CRISPRi 표적화 효율의 결정요인을 찾기 위해, 사일런싱 효율에 대한 길이, 서열 상보성 및 위치의 역할을 조사했다(도 43a). 도 40c에 제시된 바와 같이, 유전자에 따라 sgRNA 표적 서열의 위치는 효율에 중요했다. sgRNA는 mRFP 및 sfGFP 둘 다에 대한 코딩 영역의 전체 길이(sgRNA 서열에 대한 보충 데이타)를 포함하도록 추가로 설계되었다. 모든 경우에, 억제는 전사 개시 부위(도 43b)로부터 표적 거리와 역으로 서로 관련되었다. 강한 선형 상관관계가 mRFP의 경우에 관찰되었다. 유사하지만, 약간 더 약한 상관관계가 sfGFP가 표적으로 사용될 때 관찰되어 아마도 이 유전자의 스트레치에서 상이한 점 사이에 RNA 폴리머라제의 가변적인 동력학을 나타낸다.
sgRNA은 표적에 상보적인 20-bp 영역을 함유한다. 이 염기 짝짓기 영역의 중요성을 확인하기 위해, sgRNA NT1의 길이를 변경했다(도 43c). 5' 말단으로부터 영역의 확대가 사일런싱에 영향을 미치지 않는 반면, 영역의 절단은 심각하게 억제를 감소시킨다. 유전자 사일런싱에 필요한 염기 짝짓기 영역의 최소 길이는 12bp였고, 추가의 절단은 완전한 기능 손실을 유도한다. 단일 돌연변이를 sgRNA NT1의 염기 짝짓기 영역에 도입시켰고, 사일런싱에 대한 전체 효과를 시험하였다. 결과로부터, 3개의 서브-영역이 식별될 수 있고, 각각은 전체 결합 및 사일런싱에 대한 별도의 기여를 한다(도 43d). 제 1의 7 뉴클레오타이드의 임의의 단일 돌연변이는 극적으로 억제를 감소시켜 이 서열이 유형 I 및 유형 II CRISPR 시스템 둘 다에 대해 이전에 주시된 바와 같이 결합을 위한 "씨드 영역"을 구성한다는 것을 시사한다. 인접하는 뉴클레오타이드는 또한 쌍으로 돌연변이되었다(도 43e 및 도 50). 대부분의 경우에, 이중 돌연변이에 기인하는 상대적인 억제 활성은 단일 돌연변이체의 효과에 비해 증식성이어서 불일치 사이의 독립적인 관계를 시사한다. 더욱이, PAM 서열의 중요성에 대한 이전 결과와 일치하여, 부정확한 PAM은 20-bp 완전 결합 영역(도 43e)으로도 사일런싱을 완전히 파괴했다. 따라서, CRISPRi 시스템의 특이성은 PAM (2-bp) 및 적어도 12-bp sgRNA-DNA 스트레치에 의해 공동으로 결정되고, 이의 공간은 독특한 표적 부위를 위한 대부분의 박테리아 게놈을 커버하기에 충분히 크다.
모두 동일한 유전자를 표적화하는 두 개의 sgRNA를 시험하였다(도 43f 및 도 51). 다중 sgRNA의 상대적인 위치에 따라, 다른 조합 효과가 관찰되었다. 두 sgRNA를 결합시키면, 각각은 약 300-배의 억제를 갖고 증가된 전체 사일런싱을 최대 1000-배까지 허용한다. 두 개의 약한 sgRNA(약 5-배)를 결합시키면 함께 사용될 경우 증식 효과를 나타냈다. 표적이 중첩되는 두 개의 sgRNA를 사용할 때 억제성 조합 효과가 관찰되었다. 이는 아마 동일 영역에 결합하기 위한 두 sgRNA의 결쟁에 기인하였다.
도 43은 사일런싱 효율에 영향을 미치는 인자의 특성화를 묘사한다. (A) 동일한 유전자 상에서 상이한 표적화 유전자 좌(번역 개시 코돈으로부터의 거리)를 갖는 sgRNA 및 동일한 표적 유전자 좌(NT1 기반)에 대한 염기 짝짓기의 상이한 길이를 갖는 sgRNA의 사일런싱 효과를 측정하였다. (B) 사일런싱 효율은 번역 개시 코돈(오렌지 - mRFP & 그린 - sfGFP)으로부터 표적 거리와 역으로 서로 관련되었다. 상대적인 억제 활성은 최고 억제 배수 변화를 갖는 sgRNA의 억제에 대해 각 sgRNA의 억제를 표준화하여 계산했다. 오차 막대는 3개의 생물학적 복제물로부터 SEM를 나타낸다. (C) sgRNA와 표적 DNA 사이의 왓슨-크릭 염기 짝짓기의 길이는 억제 효율에 영향을 미친다. 염기 짝짓기 영역의 확대는 모두 강한 사일런싱 효과를 나타내고, 절단은 극적으로 억제를 감소시켰다. 검출가능한 억제를 위한 염기 짝짓기 영역의 최소 길이는 12-bp이다. 오차 막대는 3개의 생물학적 복제물로부터 SEM를 나타낸다. (D) 단일 불일치를 sgRNA 상의 모든 뉴클레오타이드(NT1, 도 40B)에 도입하고 이러한 단일 불일치들이 억제 효율에 영향을 미치는 방법을 측정하였다. 전체 사일런싱에 대한 별도의 중요성을 갖는 3개의 서브-영역을 식별할 수 있다. 그들은 단계 함수를 나타낸다. 제 1의 7-뉴클레오타이드 영역은 사일런싱에 중요하고, 아마 DNA 표적에 대한 sgRNA 결합을 탐침검사하기 위한 "씨드" 영역을 구성한다. PAM 서열(NGG)은 사일런싱에 필수불가결했다. 오차 막대는 3개의 생물학적 복제물로부터 SEM을 나타낸다. (E) 인접한 이중 불일치를 갖는 sgRNA의 사일런싱 효과. 단일-불일치된 sgRNA의 상대적 억제 활성은 하단부에서 표지된 불일치 위치로 제시된다. 실험적으로 측정된 이중-불일치된 sgRNA의 활성이 제시된다. 두 개의 단일-불일치된 sgRNA의 효과를 곱하여 계산된 활성은 백색으로 제시되고, "Com"으로 표지했다. 대부분의 경우에, 이중-불일치된 sgRNA의 사일런싱 활성은 간단하게 단일-불일치된 sgRNA(도 50b 제외)의 활성의 증가였고, 단일 불일치 사이의 독립적인 관계를 시사한다. 오차 막대는 3개의 생물학적 복제물의 SEM을 나타낸다. (F) 단일 mRFP 유전자를 표적화하는 이중 sgRNA을 사용하는 조합 사일런싱 효과. 동일한 유전자를 표적화하는 두 개의 sgRNA를 사용하면, 전체 녹다운 효과는 거의 1,000-배로 개선될 수 있다. 두 개의 sgRNA가 동일 유전자의 비-중첩 서열에 결합하면, 억제는 증강된다. 두 개의 sgRNA가 중첩 영역을 표적화하면, 억제는 억제된다. 오차 막대는 3개의 생물학적 복제물로부터 SEM을 나타낸다.
도 50은 도 43e에 관련되고, 인접한 이중 불일치를 갖는 sgRNA의 사일런싱 효과를 묘사한다. 단일-불일치된 sgRNA의 상대적 억제 활성은 하단부에서 표지된 불일치 위치로 제시된다. 실험적으로 측정된 이중-불일치된 sgRNA의 활성도 또한 제시된다. 두 개의 단일 불일치된 sgRNA의 효과를 곱하여 계산된 활성은 백색으로 제시되고, "Com"으로 표지된다. 형광성 결과는 3개의 생물학적 복제물의 평균 및 SEM을 나타낸다.
도 51은 도 43F에 관련되고, 단일 유전자를 조절하기 위해 두 개의 sgRNA를 사용하는 조합 사일런싱 효과를 묘사한다. 모든 경우에, 비-중첩성 sgRNA는 증가성 사일런싱 효과를 나타내고, 중첩성 sgRNA는 억제성 효과를 나타낸다. 조합 효과는 sgRNA가 템플레이트 또는 비-템플레이트 DNA 가닥을 표적화하였는지의 여부와 관계 없었다. 혈광성 결과는 3개의 생물학적 복제물의 평균 및 SEM을 나타낸다.
CRISPRi 유전자 녹다운을 사용하는 내인성 조절 네트워크의 질문
이어서, CRISPRi 시스템은 내인성 유전자 네트워크를 질문하기 위한 유전자 녹다운 플랫폼으로서 사용했다. 미생물 유전자 네트워크를 질문하는 이전 방법은 대부분 힘들고 고비용의 게놈 조작 및 녹아웃 절차에 의존한다. 반대로, CRISPRi에 의한 유전자 녹다운은 단지 목적하는 유전자에 대해 20-bp 상보적 영역을 포함하는 작은 sgRNA의 설계 및 합성을 필요로 한다. 이를 입증하기 위해, CRISPRi는 sgRNA를 충분히 특성화된 이. 콜라이 락토오스 조절 경로(도 44a)의 일부였던 유전자를 계통적으로 교란시키도록 설계하여 이. 콜라이 녹다운 균주를 생성하기 위해 사용했다. β-갈락토시다아제 검정을 수행하여 lac 억제인자 (LacI)를 억제하는 화학물질인 이소프로필 β-D-1-티오갈락토피라노사이드(IPTG)의 존재 또는 부재하에 녹다운 균주로부터 LacZ 발현을 측정하였다. 야생형 세포에서, IPTG의 부가가 LacZ 발현을 유도했다. 이 결과는 lacZ-특이적 sgRNA는 LacZ 발현(도 44b)을 강하게 억제할 수 있음을 나타냈다. 반대로, lacI 유전자를 표적화하는 sgRNA는 LacZ 발현의 직접적인 억제인자의 사일런싱이 예상되는 바와 같이, IPTG의 부재하에서도, LacZ 발현의 활성화를 유도한다.
cAMP-CRP는 프로모터의 cis 조절 부위 업스트림에 결합함으로써 LacZ 발현의 본질적인 활성제인 것으로 공지되어 있다. 지속적으로, LacZ 프로모터 중의 crp 유전자 또는 A 부위에서 표적화된 sgRNA는 억제를 유도하여 CRISPRi 실험을 사용하여 조절물질을 이의 cis-조절 서열에 결합하는 수단을 입증한다. CRP를 LacZ 프로모터에서 더욱 효과적이도록 하는 cAMP를 생산하는데 필요한 아데닐화 사이클라제 유전자(cya)의 표적화는 단지 부분적 억제를 유도했다. 성장 배지에 1mM cAMP의 부가는 crp 녹다운에 대해서가 아니라 cya 녹다운에 대한 효과를 보충하여 cya가 LacZ의 간접적인 조절물질임을 시사한다. 더욱이, sgRNA에 의한 LacI 시스-조절 부위(O 부위)의 표적화가 억제를 유도하는데, 아마도 이 부위에서 Cas9 복합체 결합이 RNA 폴리머라제를 입체적으로 차단하여 LacI 전사 억제인자의 거동을 모방하기 때문이다. 공지된 RNAP 결합 부위(P 부위)의 표적화는 또한 발현을 차단했다. 요약하면, 이러한 연구는 CRISPRi-기반 유전자 녹다운 방법은 복합체 조절 네트워크(도 44c) 중의 유전자 및 시스 요소의 조절 기능(활성화 또는 억제)을 질문하기 위한 신속하고 효과적인 접근법을 제공한다는 것을 입증한다
도 44는 CRISPRi 유전자 녹다운을 사용하는 복합체 조절 네트워크의 기능적 프로파일링을 입증한다. (A) sgRNA를 설계하고, lac 조절 경로에서 유전자(cya, crp, lacI, lacZ, lacY, lacA)를 녹다운시키거나 전사 오퍼레이터 부위(A/P/O)를 차단하는데 사용했다. LacI는 전사 오퍼레이터 부위(O 부위)에 결합시킴으로써 lacZYA 오페론의 억제인자이다. lacZ 유전자는 락토오스를 글루코스로 촉매작용을 하는 효소를 인코딩한다. 몇몇 트랜스-작용 숙주 유전자, 예를 들면, cya 및 crp는 lacZYA 시스템의 활성화에 연관된다. cAMP-CRP 복합체는 전사 오퍼레이터 부위(A 부위)에 결합하고, P 부위에 대한 RNA 폴리머라제 결합을 모집하고, 이는 lacZYA의 전사를 개시한다. LacI 기능을 억제하는 화학물질인 IPTG는 LacZ 발현을 유도한다. (B) (화이트) IPTG 부재 및 (회색) IPTG 존재하의 녹다운 균주의 β-갈락토시다아제 검정. 대조군은 CRISPRi 동요 없는 야생형 세포가 IPTG의 부가로 유도될 수 있음을 나타낸다. LacZ를 표적화하는 sgRNA는 IPTG의 존재하에서도 LacZ 발현을 강하게 억제한다. LacI가 표적화되면, LacZ 발현은 IPTG 부재하에서도 높았다. cya 및 crp 유전자의 표적화는 IPTG의 존재하에 감소된 LacZ 발현 수준을 유도했다. 1mM cAMP의 존재는 crp 녹다운이 아니라 cya 녹다운을 원조했다. 전사 오퍼레이터 부위의 차단은 LacZ 억제를 유도하여 이들이 LacZ에 대한 중요한 시스-작용 조절 부위임을 시사한다. 동요시, LacZ의 감소된(하향 화살표) 및 증가된(상향 화살표) 발현이 명시된다. 오차 막대는 3개의 생물학적 복제물로부터 SEM을 나타낸다. (C) 녹다운 실험은 lac 실험실 회로에서 조절물질의 역할을 프로파일링할 수 있도록 한다. 데이타는 IPTG 없이 LacZ 활성을 나타내는 x-축과 IPTG와 함께 이의 활성을 나타내는 y-축을 갖는 2-D 그래프에 나타낸다. 각 축을 따라 타원형의 확산은 표준 편차를 나타낸다. β-갈락토시다아제 검정 결과는 3개의 생물학적 복제물의 평균 및 SEM을 나타낸다. LacI 및 LacZ 표적화에 대한 RNA-seq 데이타는 또한 도 49를 참조한다.
CRISPRi는 인간 세포에서 표적화된 유전자 발현을 녹다운시킬 수 있다
전사를 억제하기 위해 dCas9-sgRNA 복합체를 사용하는 CRISPRi 접근법의 일반성을 시험하기 위해, 시스템을 HEK293 포유동물 세포에서 시험하였다. dCas9 단백질은 코돈 최적화하고, 핵 국재화 서열(NLS)의 3개의 카피에 융합시키고, 뮤린 줄기세포 바이러스(MSCV) 레트로바이러스 벡터로부터 발현시켰다. 도 40b에 도시된 동일한 sgRNA 설계를 사용하여 RNA 폴리머라제 III U6 프로모터로부터 발현시켰다. SV40 프로모터 하에 EGFP를 발현시키는 리포터 HEK293 세포주는 바이러스 감염에 의해 생성시켰다. EGFP 코딩 영역의 비-템플레이트 DNA 가닥을 표적화한 sgRNA(eNT2)를 사용하여, 유전자 발현의 적당하지만 재생가능한 녹다운을 관찰하였다(46% 억제, 도 45a). 억제는 dCas9 단백질 및 sgRNA 둘다에 의존하여 억제가 dCas9-sgRNA 복합체 및 RNA-유도된 표적화에 기인하였음을 의미하였다. 동일한 sgRNA는 플라스미드로부터 일시적으로 발현될 때, 동일 유전자에 대해 우수한 발현을 나타냈다(63% 억제, 도 52). 박테리아 시스템과 일치하여, 비-템플레이트 가닥에 표적화된 sgRNA 만이 억제를 나타냈다. 전사 개시로부터의 거리 및 국소 염색질 상태와 같은 인자는 억제 효율(도 52)을 결정하는 중요한 파라미터일 수 있다. dCas9 및 sgRNA 발현, 안정성, 핵 국재화, 및 상호작용의 최적화는 포유동물 세포에서 CRISPRi 효율의 추가 향상을 허용한다.
도 45는 CRISPRi가 인간 세포에서 유전자 발현을 억제할 수 있음을 입증한다. (A) HEK293 세포 중의 CRISPRi 시스템. SV40-EGFP 발현 카세트를 레트로바이러스 감염을 통해 게놈에 삽입시켰다. dCas9 단백질을 코돈-최적화하고, NLS 서열의 3개 카피에 융합시켰다. sgRNA는 RNA 폴리머라제 III U6 벡터로부터 발현시켰다. EGFP의 비-템플레이트 가닥을 표적화하는 dCas9 및 sgRNA(eNT2)의 공동-형질감염은 형광성을 감소시키고(약 46%), 반면 dCas9 또는 sgRNA 단독의 발현은 어떤 효과도 나타내지 않았다. (B) dCas9:sgRNA-매개된 억제는 표적 유전자 좌에 의존했다. 7개의 sgRNA는 템플레이트 또는 비-템플레이트 가닥 상의 EGFP 코딩 서열의 상이한 영역을 표적화하도록 설계되었다. 단지 eNT2 및 eNT5가 적당한 억제를 나타냈다. 7A 및 7B로부터의 형광성 결과는 2개의 생물학적 복제물의 평균 및 오차를 나타낸다.
도 52는 도 45와 관련되고, sgRNA 억제는 표적 유전자 좌 및 비교적 전사 개시로부터의 거리에 의존한다는 것을 나타낸다. 동일한 sgRNA를 사용하여 상이한 프로모터를 갖는 동일한 EGFP 유전자를 억제하였다. Cas9/sgRNA 복합체는 일시적으로 형질감염된 플라스미드 DNA로부터의 전사를 억제한다. 전사 억제 수준은 게놈 유전자에 대해 관찰된 것보다 약간 우수했고(63%), GFP-음성 세포의 퍼센트는 sgRNA의 존재하에 증가되었다. 표적 유전자 좌는 전사 개시로부터 상이한 거리를 갖는다. SV40-EGFP가 억제를 나타내는 반면, LTR-EGFP는 어떤 효과도 갖지 않았다. 형광성 결과는 2개의 생물학적 복제물의 평균 및 오차를 나타낸다.
CRISPRi는 표적 유전자의 전사를 효율적으로 및 선택적으로 억제한다
CRISPRi 시스템은 표적화된 유전자 조절을 위한 비교적 간단한 플랫폼이다. CRISPRi는 복합체 숙주 인자의 존재에 의존하지 않지만, 대신 단지 dCas9 단백질 및 가이드 RNA를 필요로 하고, 따라서 가요성이고 매우 설계가능하다. 상기 시스템은 박테리아 중 유전자를 효율적으로 침묵시킬 수 있다. 사일런싱은, 부정확한 효과가 검출되지 않았기 때문에, 매우 효율적이다. 더욱이, 녹다운의 효율은 표적 유전자 좌 및 sgRNA와 표적 유전자 사이의 염기 짝짓기 정도를 변화시켜 조정할 수 있다. 이는 필수적인 유전자의 연구에 특히 유용한 특징-- 하이포모르프의 대립유전자 시리즈를 생성시킬 수 있게 한다. 상기 시스템은 sgRNA를 설계함으로써 용이하게 프로그래밍될 수 있는 방식으로 전사를 직접 차단함으로써 기능한다. 기계론적으로, 이것은 RNAi-기반 사일런싱과 다르고, 이는 이미 전사된 mRNA의 파괴를 필요로 한다.
또한, 이러한 dCas9:sgRNA 복합체는 또한 임의의 프로모터 내에서 중요한 시스-작용 모티프를 표적화하고, 그들의 동족 트랜스-작용 전사 인자의 회합을 입체적으로 차단하여 전사를 조절할 수 있다. 따라서, 유전자 녹다운 도구로서의 이의 용도 이외에, CRISPRi는 프로모터와 기타 게놈 조절 모듈의 기능적 맵핑에 사용될 수 있었다.
CRISPRi는 게놈-규모 분석 및 조절에 따른다
CRISPRi 방법은 DNA-표적화 RNA의 사용을 기반으로 하고, 단지 DNA 표적화 세그먼트는 특이적 유전자 표적을 위해 설계될 필요가 있다. 대규모 DNA 올리고뉴클레오타이드 합성 기술의 진보로, 게놈 표적화를 위한 독특한 20-bp 영역을 함유하는 올리고뉴클레오타이드의 대규모 세트를 생성하는 것은 신속하고 저렴하다. 이러한 올리고뉴클레오타이드 라이브러리는 유전자 기능을 추론하기 위해 개별적 유전자 다수를 표적화하거나 유전적 상호작용을 맵핑하기 위해 유전자 쌍을 표적화하도록 할 수 있었다. 더욱이, CRISPRi는, 작은 크기의 sgRNA가 복수 요소를 동일한 발현 벡터에 연결하도록 하기 때문에, 큰 세트의 유전자의 발현을 동시에 조절하는데 사용할 수 있었다.
CRISPRi는 미생물 게놈을 조작하기 위한 새로운 도구를 제공한다
CRISPRi 플랫폼이 조밀하고 자급식이기 때문에, 이는 상이한 유기체에 적응될 수 있다. CRISPRi는 병원균 또는 산업적으로 유용한 유기체를 포함하여, 유전공학 방법이 충분히 개발되지 않은 비-모델 유기체를 연구하기 위한 강력한 도구이다. 대부분의 진핵생물과 달리, 대부분의 박테리아는 RNAi 기구가 부족하다. 그 결과, 설계된 합성 RNA을 사용하는 내인성 유전자의 조절은 현재 제한된다. CRISPRi는 미생물의 유전자 동요를 위한 RNAi-형 방법을 제공할 수 있다.
전사 조절 네트워크를 조작하기 위한 플랫폼으로서 CRISPRi
CRISPRi는 전사 조절 네트워크를 조작하기 위한 가요성 뼈대로서 사용할 수 있다. CRISPRi 플랫폼은, 본질적으로 RNA-유도된 DNA-결합 복합체이기 때문에, 또한 게놈 내의 특이적 부위에 다양한 조절 기구를 지시하기 위한 가요성 스캐폴드를 제공한다. 표적 유전자의 전사를 단순히 차단함을 넘어, 상이한 생물학적 과정을 조정하고 상이한 기능적 결과(즉, 전사 활성화, 염색질 변형)을 생성하기 위해 dCas9 단백질을 다수의 조절 도메인과 결합시킬 수 있다.
CRISPRi 시스템에서, 다중 sgRNA를 하나의 업스트림 sgRNA가 상이한 다운스트림 sgRNA의 발현을 조절하는 전사 회로에 연결할 수 있다. 미생물 중의 RNA 분자는 단명하는 경향이 있기 때문에, 본 발명자들은 sgRNA에 의해 조절된 유전적 프로그램이 단백질 발현 및 분해와 같은 느린 공정을 수반하는 회로와 상이한 신속한 동력학을 나타낼 것으로 추측한다. 요약하면, CRISPRi 시스템은 다양한 생체의학 연구 및 게놈-규모 기능적 프로파일링, 미생물 대사 공학 및 세포 재프로그래밍을 포함한 임상 적용에 적합한 일반적인 유전적 프로그램밍 플랫폼이다.
실시예 5: 키메라 부위 지향적 폴리펩타이드는 인간 세포에서 전사를 조절(활성화 또는 억제)하기 위해 사용될 수 있다.
본 발명자들은 인간 세포에서, 촉매적으로 불활성인 Cas9를 포함하는 융합 단백질 및 활성제 도메인 또는 억제인자 도메인은 각각 표적 DNA로부터의 전사를 증가시키거나 감소시킬 수 있음을 입증했다.
도 55. 본 발명자들은 인간화된 촉매적으로 불활성 Cas9를 전사 활성제 도메인 VP64와 융합시켰다. (A) 이 시스템을 사용하여 유전자 활성화 효율을 시험하기 위해, 본 발명자들은 GFP를 조절하는 GAL4 UAS 유도성 프로모터를 HEK293(인간 조직 배양 세포) 게놈에 삽입했다. (B) GAL4 UAS 프로모터는 효모-유도된 단백질 GAL4의 존재하에 유도될 수 있다. dCas9-VP64 융합은 GAL4 UAS 영역에 결합하는 동족 가이드 RNA의 존재하에 GAL4 UAS를 20-배까지 효과적으로 활성화시킬 수 있다. (C) dCas9-VP64 활성화를 위한 현미경적 이미지. (D) dCas9-VP64 활성화를 위한 유세포측정 데이타.
도 56 본 발명자들은 인간화된 촉매적으로 불활성 Cas9를 전사 억제인자 도메인 KRAB과 융합시켰다. (상단부) 본 발명자들은 충분히 특성화된 프로모터 SV40 조기 프로모터를 표적화하는 10 가이드 RNA 및 EGFP 코딩 영역을 표적화하는 하나의 가이드 RNA를 설계했다. (하단부) 비-키메라 dCas9를 사용하여, 본 발명자들은 P9 및 NT2의 gRNA에 대해 2 내지 3배 발현을 관찰했다. 이 효율은 dCas9-KRAB 융합을 사용하여 크게 향상되었다. 예를 들면, dCas9-KRAB 융합으로, P9 및 NT2는 각각 20-배 및 15-배 억제를 나타냈다. 또한, P1-P6은 융합 단백질이 사용될 때 발현의 상당한 감소를 나타내고, 비-키메라 dCas9가 사용될 때 제한된 억제를 나타냈다.
실시예 6: Cas9는 표적 DNA 절단을 수행하기 위해 자연에 존재하지 않는 인공 가이드 RNA를 사용할 수 있다
인공 crRNA 및 인공 tracrRNA는 에스. 파이오제네스 crRNA 및 tracrRNA의 자연 발생 전사체의 단백질 결합 세그먼트에 기초하여 설계하고, 천연 에스. 파이오제네스 crRNA:tracrRNA 듀플렉스 내의 비대칭 돌출(도 57a에 묘사된 인공(상단부) 및 천연(하단부) RNA 분자 둘 다의 단백질 결합 도메인 중의 돌출)을 모방하기 위해 변형했다. 인공 tracrRNA 서열은 천연 tracrRNA과 50% 미만 동일성을 공유한다. crRNA:tracrRNA 단백질-결합 듀플렉스의 예상된 2차 구조는 두 RNA 쌍에 대해 동일하지만, RNA 나머지의 예상된 구조가 훨씬 상이하다.
도 57은 자연 발생 tracrRNAs 및 crRNA와 매우 적게(거의 50% 동일성) 공유하는 인공 서열이 DNA-표적화 RNA의 단백질 결합 도메인의 구조가 보존되는 한 표적 DNA를 절단하도록 Cas9로 기능할 수 있음을 입증한다. (A) 에스 . 파이오제네스 tracrRNA 및 crRNA 및 인공 tracrRNA 및 crRNA의 공동-폴딩. (B) 에스 . 파이오제네스 Cas9 및 tracrRNA:crRNA 오쏘로그의 조합을 사용하여 플라스미드 DNA 절단 검정을 수행했다. Spy - 에스 . 파이오제네스, Lin - 엘. 이노쿠아, Nme - 엔. 메닌자 이티디스, Pmu - 피. 멀토시다. 에스 . 파이오제네스 Cas9는 선택된 박테리아 종 중의 자연 발생 tracrRNA:crRNA 오쏘로그 모두가 아니라 일부에 의해 가이드될 수 있다. 현저하게, 에스. 파이오제네스 Cas9는 인공 tracrRNA:crRNA 쌍에 의해 가이드될 수 있고, 이는 CRISPR 시스템과 완전히 관련되지 않는 서열을 사용하는 자연 발생 DNA-표적화 RNA의 단백질 결합 세그먼트의 구조를 기반으로 설계했다.
사용된 인공 "tracrRNA"(활성제 RNA)는 5'-GUUUUCCCUUUUCAAAGAAAUCUCCUGGGCACCUAUCUUCUUAGGUGCCCUCCCUUGUUUAAACCUGACCAGUUAACCGGCUGGUUAGGUUUUU-3'(서열번호 1347)였다. 사용된 인공 "crRNA"(타겟터 RNA)는 5'-GAGAUUUAUGAAAAGGGAAAAC-3'(서열번호 1348)였다.
실시예 7: 비-인간 이식유전자 유기체의 발생
Cas9를 발현하는 이식유전자 마우스(비변형된, 감소된 효소 활성을 갖도록 변형된, 상기 개요된 임의의 목적을 위해 융합 단백질로서 변형된)는 당해 기술 분야의 숙련가에게 공지된 편리한 방법(예를 들면, (i) 마우스 배아 줄기 세포(ES 세포)의 표적화된 유전자 좌(예: ROSA 26)에서 유전자 녹-인 후 배반포 주입 및 키메라 마우스의 생성; (ii) 랜덤하게 통합된 이식유전자의 수정된 마우스 난모세포의 전핵으로의 주입에 이어, 가임신 여성에게 난자 이식; 등)을 사용하여 생성된다. Cas9 단백질은 적어도 배아 줄기 세포에서 발현하는 프로모터의 제어하에 존재하고, 추가로 일시적 또는 조직-특이적 조절(예를 들면, 약물 유도성, Cre/Lox 기반 프로모터 시스템에 의해 조절됨, 등)하에 존재할 수 있다. 이식유전자 Cas9 발현 마우스의 라인이 생성되면, 배아 줄기 세포를 단리하여 배양하고, 일부 경우에서 ES 세포는 미래의 사용을 위해 냉동시킨다. 단리된 ES 세포가 Cas9를 발현시키기 때문에(및 일부 경우에서 발현은 일시적 제어하(예: 약물 유도성)에 존재한다), 새로운 녹-아웃 또는 녹-인 세포 (및 따라서 마우스)는 Cas9를 표적화하는 적당하게 설계된 DNA-표적화 RNA를 선택된 특별한 유전자 좌에 도입함으로써 게놈 중의 임의의 목적하는 유전자 좌에서 신속하게 생성된다. 이러한 시스템 및 이의 다수의 변형체를 사용하여 선택된 임의의 유전자 좌에서 새로운 유전적으로 변형된 유기체를 생성한다. 변형된 Cas9가 전사를 조절하고/하거나 DNA를 변형시키고/시키거나 DNA와 관련된 폴리펩타이드를 변형시키기 위해 사용될 경우, ES 세포 자체 (또는 ES 세포(예를 들면, 전체 마우스, 분화된 세포주 등)로부터 유도된 임의의 분화된 세포는 적합하게 DNA-표적화 RNA를 목적하는 Cas9 발현 세포에 도입함으로써 간단히 선택된 임의의 유전자 (또는 선택된 임의의 발현 생성물, 또는 선택된 임의의 게놈 유전자 좌)의 특성을 연구하기 위해 사용된다.
본 발명은 이의 특정 구현예를 참조로 기술되었지만, 다양한 변화가 수행될 수 있고, 등가물이 본 발명의 진정한 취지 및 범위로부터 벗어나지 않고 치환될 수 있음이 당해 기술 분야의 숙련가에 의해 이해되어야 한다. 또한, 특정 상황, 물질, 물질의 조성물, 공정, 공정 단계 또는 단계들을 본 발명의 목적, 정신 및 범위에 적응시키기 위해 많은 변형이 수행될 수 있다. 이러한 변형 모두는 여기에 첨부된 특허청구범위의 범위 내에 속하는 것으로 의도된다.
Claims (155)
- 하기를 포함하는 DNA-표적화 RNA:
(i) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및
(ii) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절. - 청구항 1에 있어서, 상기 제 1 분절은 표적 DNA에서 서열에 대해 100% 상보성을 갖는 8 개의 뉴클레오타이드를 포함하는, DNA-표적화 RNA.
- 청구항 1 에 있어서, 상기 제 2 분절은 적어도 8 개의 인접 뉴클레오타이드의 스트레치에 대해 서열번호:563-682에서 제시된 뉴클레오타이드 서열의 임의의 하나와 적어도 60% 동일성을 갖는 뉴클레오타이드 서열, 또는 그의 보체를 포함하는, DNA-표적화 RNA.
- 청구항 1 에 있어서, 상기 제 2 분절은 적어도 8 개의 인접 뉴클레오타이드의 스트레치에 대해 서열번호:431-562에서 제시된 뉴클레오타이드 서열의 임의의 하나와 적어도 60% 동일성을 갖는 뉴클레오타이드 서열, 또는 그의 보체를 포함하는, DNA-표적화 RNA.
- 청구항 1에 있어서, 상기 부위 지향적 변형 폴리펩타이드는 도 3에서 묘사된 Cas9/Csn1 아미노산 서열의 아미노산 7-166 또는 731-1003, 또는 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 상응하는 부분과 적어도 약 75% 아미노산 서열 동일성을 갖는 아미노산 서열을 포함하는, DNA-표적화 RNA.
- 청구항 1의 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는, DNA 폴리뉴클레오타이드.
- 청구항 6의 DNA 폴리뉴클레오타이드를 포함하는, 재조합 발현 벡터.
- 청구항 7에 있어서, 상기 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열은 프로모터에 작동가능하게 연결되는, 재조합 발현 벡터.
- 청구항 8에 있어서, 상기 프로모터는 유도성 프로모터인 재조합 발현 벡터.
- 청구항 7에 있어서, 상기 청구항 1의 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열은 다중 클로닝 부위를 추가로 포함하는, 재조합 발현 벡터.
- 청구항 6의 DNA 폴리뉴클레오타이드를 포함하는 시험관내 유전적으로 변형된 숙주세포.
- 하기를 포함하는 재조합 발현 벡터:
(i) 하기를 포함하는 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열:
(a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및
(b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절; 및
(ii) 하기를 포함하는 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열:
(a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및
(b) 부위 지향적 효소 활성을 나타내는 활성부로서, 상기 효소 활성의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부. - 하기를 포함하는 재조합 발현 벡터:
(i) 하기를 포함하는 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열:
(a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및
(b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절; 및
(ii) 하기를 포함하는 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열:
(a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및
(b) 표적 DNA 내에서 전사를 조절하는 활성부로서, 상기 표적 DNA 내의 조절된 전사의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부. - 하기를 포함하는 변이체 부위 지향적 변형 폴리펩타이드:
(i) DNA-표적화 RNA와 상호작용하는 RNA-결합부로서, 상기 DNA-표적화 RNA는 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 결합부; 및
(ii) 감소된 부위 지향적 효소 활성를 나타내는 활성부로서, 상기 효소 활성의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부. - 청구항 14에 있어서, 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 S. 파이오제네스 서열 서열번호:8의 H840A 돌연변이 또는 상응하는 돌연변이를 포함하는, 변이체 부위 지향적 변형 폴리펩타이드.
- 청구항 14에 있어서, 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 S. 파이오제네스 서열 서열번호:8의 D10A 돌연변이 또는 상응하는 돌연변이를 포함하는, 변이체 부위 지향적 변형 폴리펩타이드.
- 청구항 14에 있어서, (i) 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 S. 파이오제네스 서열 서열번호:8의 D10A 돌연변이 또는 상응하는 돌연변이; 및 (ii) 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 S. 파이오제네스 서열 서열번호:8의 H840A 돌연변이 또는 상응하는 돌연변이 둘 모두를 포함하는, 변이체 부위 지향적 변형 폴리펩타이드.
- 하기를 포함하는 키메라 부위 지향적 변형 폴리펩타이드:
(i) DNA-표적화 RNA와 상호작용하는 RNA-결합부로서, 상기 DNA-표적화 RNA는 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 결합부; 및
(ii) 부위 지향적 효소 활성을 나타내는 활성부로서, 상기 효소 활성의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부. - 청구항 18에 있어서, 도 3에서 묘사된 Cas9/Csn1 아미노산 서열의 아미노산 7-166 또는 731-1003, 또는 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 상응하는 부분과 적어도 약 75% 아미노산 서열 동일성을 갖는 아미노산 서열을 포함하는, 키메라 부위 지향적 변형 폴리펩타이드.
- 청구항 18에 있어서, 상기 DNA-표적화 RNA는 적어도 8 개의 인접 뉴클레오타이드의 스트레치에 대해 서열번호:563-682에서 제시된 뉴클레오타이드 서열의 임의의 하나와 적어도 60% 동일성을 갖는 뉴클레오타이드 서열을 추가로 포함하는, 키메라 부위 지향적 변형 폴리펩타이드.
- 청구항 18에 있어서, 상기 DNA-표적화 RNA는 적어도 8 개의 인접 뉴클레오타이드의 스트레치에 대해 서열번호:431-562에서 제시된 뉴클레오타이드 서열의 임의의 하나와 적어도 60% 동일성을 갖는 뉴클레오타이드 서열을 추가로 포함하는, 키메라 부위 지향적 변형 폴리펩타이드.
- 청구항 18에 있어서, 상기 효소 활성은 표적 DNA를 변형시키는, 키메라 부위 지향적 변형 폴리펩타이드.
- 청구항 22에 있어서, 상기 효소 활성은 뉴클레아제 활성, 메틸전이효소 활성, 탈메틸효소 활성, DNA 치유 활성, DNA 손상 활성, 탈아미노화 활성, 디스무타제 활성, 알킬화 활성, 탈퓨린화 활성, 산화 활성, 피리미딘 이량체 형성 활성, 인테그라제 활성, 전위효소 활성, 재조합효소 활성, 폴리머라제 활성, 리가제 활성, 헬리카제 활성, 광분해효소 활성 또는 글리코실라제 활성인, 키메라 부위 지향적 변형 폴리펩타이드.
- 청구항 23에 있어서, 상기 효소 활성은 뉴클레아제 활성인, 키메라 부위 지향적 변형 폴리펩타이드.
- 청구항 24에 있어서, 상기 뉴클레아제 활성은 표적 DNA에서 이중 가닥 절단을 도입하는, 키메라 부위 지향적 변형 폴리펩타이드.
- 청구항 18에 있어서, 상기 효소 활성은 표적 DNA와 연관된 표적 폴리펩타이드를 변형시키는, 키메라 부위 지향적 변형 폴리펩타이드.
- 청구항 26에 있어서, 상기 효소 활성은 메틸전이효소 활성, 탈메틸효소 활성, 아세틸전이효소 활성, 탈아세틸화효소 활성, 키나아제 활성, 포스파타제 활성, 우비퀴틴 리가제 활성, 탈우비퀴틴화 활성, 아데닐화 활성, 탈아데닐화 활성, 스모화 활성, 탈스모화 활성, 리보실화 활성, 탈리보실화 활성, 미리스토일화 활성 또는 탈미리스토일화 활성인, 키메라 부위 지향적 변형 폴리펩타이드.
- 청구항 26에 있어서, 상기 표적 폴리펩타이드는 히스톤이고 상기 효소 활성은 메틸전이효소 활성, 탈메틸효소 활성, 아세틸전이효소 활성, 탈아세틸화효소 활성, 키나아제 활성, 포스파타제 활성, 우비퀴틴 리가제 활성 또는 탈우비퀴틴화 활성인, 키메라 부위 지향적 변형 폴리펩타이드.
- 청구항 18의 키메라 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 RNA 폴리뉴클레오타이드.
- 청구항 18의 키메라 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는, DNA 폴리뉴클레오타이드.
- 청구항 30의 폴리뉴클레오타이드를 포함하는, 재조합 발현 벡터.
- 청구항 31에 있어서, 상기 폴리뉴클레오타이드는 프로모터에 작동가능하게 연결되는, 재조합 발현 벡터.
- 청구항 32에 있어서, 상기 프로모터는 유도성 프로모터인 재조합 발현 벡터.
- 청구항 30의 폴리뉴클레오타이드를 포함하는, 시험관내에서 유전적으로 변형된 숙주세포.
- 하기를 포함하는 키메라 부위 지향적 변형 폴리펩타이드:
(i) DNA-표적화 RNA와 상호작용하는 RNA-결합부로서, 상기 DNA-표적화 RNA는 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 결합부; 및
(ii) 표적 DNA 내에서 전사를 조절하는 활성부로서, 상기 표적 DNA 내의 조절된 전사의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부. - 청구항 35에 있어서, 상기 활성부는 표적 DNA 내에서 전사를 증가시키는, 키메라 부위 지향적 변형 폴리펩타이드.
- 청구항 35에 있어서, 상기 활성부는 표적 DNA 내에서 전사를 감소시키는, 키메라 부위 지향적 변형 폴리펩타이드.
- DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및 부위 지향적 효소 활성을 나타내는 활성부로서, 상기 효소 활성의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부를 포함하는 재조합 부위 지향적 변형 폴리펩타이드를 포함하는, 유전적으로 변형된 세포.
- 청구항 38에 있어서, 상기 부위 지향적 변형 폴리펩타이드는 도 3에서 묘사된 Cas9/Csn1 아미노산 서열의 아미노산 7-166 또는 731-1003과, 또는 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 상응하는 부분과 적어도 약 75% 아미노산 서열 동일성을 갖는 아미노산 서열을 포함하는, 유전적으로 변형된 세포.
- 청구항 38에 있어서, 상기 세포는 하기로 이루어진 그룹으로부터 선택되는, 유전적으로 변형된 세포: 고세균 세포, 박테리아 세포, 진핵 세포, 진핵 단일-세포 유기체, 체세포, 생식세포, 줄기세포, 식물 세포, 조류 세포, 동물 세포, 무척추동물 세포, 척추동물 세포, 물고기 세포, 개구리 세포, 새 세포, 포유동물 세포, 돼지 세포, 암소 세포, 염소 세포, 양 세포, 설치류 세포, 랫트 세포, 마우스 세포, 비-인간 영장류 세포, 및 인간 세포.
- 게놈이 하기를 포함하는 재조합 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 이식유전자를 포함하는 이식유전자 비-인간 유기체: (i) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및 (ii) 부위 지향적 효소 활성을 나타내는 활성부로서, 상기 효소 활성의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부.
- 청구항 41에 있어서, 상기 부위 지향적 변형 폴리펩타이드는 도 3에서 묘사된 Cas9/Csn1 아미노산 서열의 아미노산 7-166 또는 731-1003과, 또는 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 상응하는 부분과 적어도 약 75% 아미노산 서열 동일성을 갖는 아미노산 서열을 포함하는 이식유전자 유기체.
- 청구항 41에 있어서, 상기 유기체는 하기로 이루어진 그룹으로부터 선택되는 이식유전자 유기체: 고세균, 박테리아, 진핵 단일-세포 유기체, 조류, 식물, 동물, 무척추동물, 파리, 벌레, 자포동물, 척추동물, 물고기, 개구리, 새, 포유동물, 유제류, 설치류, 랫트, 마우스, 및 비-인간 영장류.
- 하기를 포함하는 조성물:
(i) 하기를 포함하는 DNA-표적화 RNA, 또는 이것을 인코딩하는 DNA 폴리뉴클레오타이드:
(a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및
(b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절; 및
(ii) 하기를 포함하는 부위 지향적 변형 폴리펩타이드, 또는 그것을 인코딩하는 폴리뉴클레오타이드:
(a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및
(b) 부위 지향적 효소 활성을 나타내는 활성부로서, 상기 효소 활성의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부. - 청구항 44에 있어서, 상기 DNA-표적화 RNA의 제 1 분절은 표적 DNA에서 서열에 대해 적어도 100% 상보적인 8 개의 뉴클레오타이드를 포함하는, 조성물.
- 청구항 44에 있어서, 상기 DNA-표적화 RNA의 제 2 분절은 적어도 8 개의 인접 뉴클레오타이드의 스트레치에 대해 서열번호:563-682에서 제시된 뉴클레오타이드 서열의 임의의 하나와 적어도 60% 동일성을 갖는 뉴클레오타이드 서열을 포함하는, 조성물.
- 청구항 44에 있어서, 상기 DNA-표적화 RNA의 제 2 분절은 적어도 8 개의 인접 뉴클레오타이드의 스트레치에 대해 서열번호:431-562에서 제시된 뉴클레오타이드 서열의 임의의 하나와 적어도 60% 동일성을 갖는 뉴클레오타이드 서열을 포함하는, 조성물.
- 청구항 44에 있어서, 상기 부위 지향적 변형 폴리펩타이드는 도 3에서 묘사된 Cas9/Csn1 아미노산 서열의 아미노산 7-166 또는 731-1003과, 또는 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 상응하는 부분과 적어도 약 75% 아미노산 서열 동일성을 갖는 아미노산 서열을 포함하는, 조성물.
- 청구항 44에 있어서, 상기 효소 활성은 표적 DNA를 변형시키는, 조성물.
- 청구항 49에 있어서, 상기 효소 활성은 뉴클레아제 활성, 메틸전이효소 활성, 탈메틸효소 활성, DNA 치유 활성, DNA 손상 활성, 탈아미노화 활성, 디스무타제 활성, 알킬화 활성, 탈퓨린화 활성, 산화 활성, 피리미딘 이량체 형성 활성, 인테그라제 활성, 전위효소 활성, 재조합효소 활성, 폴리머라제 활성, 리가제 활성, 헬리카제 활성, 광분해효소 활성 또는 글리코실라제 활성인, 조성물.
- 청구항 50에 있어서, 상기 효소 활성은 뉴클레아제 활성인, 조성물.
- 청구항 51 에 있어서, 상기 뉴클레아제 활성은 표적 DNA에서 이중 가닥 절단을 도입하는, 조성물.
- 청구항 44에 있어서, 상기 효소 활성은 표적 DNA와 연관된 표적 폴리펩타이드를 변형시키는, 조성물.
- 청구항 53에 있어서, 상기 효소 활성은 메틸전이효소 활성, 탈메틸효소 활성, 아세틸전이효소 활성, 탈아세틸화효소 활성, 키나아제 활성, 포스파타제 활성, 우비퀴틴 리가제 활성, 탈우비퀴틴화 활성, 아데닐화 활성, 탈아데닐화 활성, 스모화 활성, 탈스모화 활성, 리보실화 활성, 탈리보실화 활성, 미리스토일화 활성 또는 탈미리스토일화 활성인, 조성물.
- 청구항 53에 있어서, 상기 표적 폴리펩타이드는 히스톤이고 상기 효소 활성은 메틸전이효소 활성, 탈메틸효소 활성, 아세틸전이효소 활성, 탈아세틸화효소 활성, 키나아제 활성, 포스파타제 활성, 우비퀴틴 리가제 활성 또는 탈우비퀴틴화 활성인, 조성물.
- 청구항 44에 있어서, 상기 DNA-표적화 RNA는 이중-분자 DNA-표적화 RNA이고 상기 조성물은 타겟터-RNA 및 활성제-RNA 둘 모두를 포함하고, 그것의 듀플렉스-형성 분절은 상보적이고 혼성화되어 DNA-표적화 RNA의 제 2 분절을 형성하는, 조성물.
- 청구항 56에 있어서, 상기 활성제-RNA의 듀플렉스-형성 분절은 적어도 8 개의 인접 뉴클레오타이드의 스트레치에 대해 서열번호:431-682에서 제시된 뉴클레오타이드 서열의 임의의 하나와 적어도 60% 동일성을 갖는 뉴클레오타이드 서열을 포함하는, 조성물.
- 하기를 포함하는 조성물:
(i) 청구항 44의 DNA-표적화 RNA, 또는 그것을 인코딩하는 DNA 폴리뉴클레오타이드; 및
(ii) 핵산을 안정화시키는 완충제. - 하기를 포함하는 조성물:
(i) 청구항 44의 부위 지향적 변형 폴리펩타이드, 또는 그것을 인코딩하는 폴리뉴클레오타이드; 및
(ii) 핵산 및/또는 단백질을 안정화시키는 완충제. - 하기를 포함하는 조성물:
(i) 하기를 포함하는 DNA-표적화 RNA, 또는 이것을 인코딩하는 DNA 폴리뉴클레오타이드:
(a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및
(b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절; 및
(ii) 하기를 포함하는 부위 지향적 변형 폴리펩타이드, 또는 그것을 인코딩하는 폴리뉴클레오타이드:
(a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및
(b) 표적 DNA 내에서 전사를 조절하는 활성부로서, 상기 표적 DNA 내의 조절된 전사의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부. - 청구항 60에 있어서, 상기 활성부는 표적 DNA 내에서 전사를 증가시키는, 조성물.
- 청구항 60에 있어서, 상기 활성부는 표적 DNA 내에서 전사를 감소시키는, 조성물.
- 하기를 포함하는 조성물:
(i) 청구항 60의 부위 지향적 변형 폴리펩타이드, 또는 그것을 인코딩하는 폴리뉴클레오타이드; 및
(ii) 핵산 및/또는 단백질을 안정화시키는 완충제. - 표적 DNA를 하기와 접촉시키는 단계를 포함하는, 표적 DNA의 부위 특이적인 변형 방법:
(i) 하기를 포함하는 DNA-표적화 RNA, 또는 이것을 인코딩하는 DNA 폴리뉴클레오타이드:
(a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및
(b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절; 및
(ii) 하기를 포함하는 부위 지향적 변형 폴리펩타이드, 또는 그것을 인코딩하는 폴리뉴클레오타이드:
(a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및
(b) 부위 지향적 효소 활성을 나타내는 활성부. - 청구항 64에 있어서, 상기 표적 DNA는 염색체외인 것인, 방법.
- 청구항 64에 있어서, 상기 표적 DNA는 5'-CCY-3'인 상보적 가닥의 PAM 서열을 포함하고, 여기서 Y는 임의의 DNA 뉴클레오타이드이고 Y는 표적 DNA의 상보적 가닥의 표적 서열의 5' 바로 옆에 있는 것인, 방법.
- 청구항 64에 있어서, 상기 표적 DNA는 시험관 내의 염색체의 부분인, 방법.
- 청구항 64 에 있어서, 상기 표적 DNA는 생체 내의 염색체의 부분인, 방법.
- 청구항 64에 있어서, 상기 표적 DNA는 세포 중 염색체의 부분인, 방법.
- 청구항 69에 있어서, 상기 세포는 하기로 이루어진 그룹으로부터 선택되는 방법: 고세균 세포, 박테리아 세포, 진핵 세포, 진핵 단일-세포 유기체, 체세포, 생식세포, 줄기세포, 식물 세포, 조류 세포, 동물 세포, 무척추동물 세포, 척추동물 세포, 물고기 세포, 개구리 세포, 새 세포, 포유동물 세포, 돼지 세포, 암소 세포, 염소 세포, 양 세포, 설치류 세포, 랫트 세포, 마우스 세포, 비-인간 영장류 세포, 및 인간 세포.
- 청구항 64에 있어서, 상기 DNA-표적화 RNA는 적어도 8 개의 인접 뉴클레오타이드의 스트레치에 대해 서열번호:563-682에서 제시된 뉴클레오타이드 서열의 임의의 하나와 적어도 60% 동일성을 갖는 뉴클레오타이드 서열을 포함하는, 방법.
- 청구항 64에 있어서, 상기 DNA-표적화 RNA는 적어도 8 개의 인접 뉴클레오타이드의 스트레치에 대해 서열번호:431-562에서 제시된 뉴클레오타이드 서열의 임의의 하나와 적어도 60% 동일성을 갖는 뉴클레오타이드 서열을 포함하는, 방법.
- 청구항 64에 있어서, 상기 DNA-변형 폴리펩타이드는 도 3에서 묘사된 Cas9/Csn1 아미노산 서열의 아미노산 7-166 또는 731-1003과, 또는 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 상응하는 부분과 적어도 약 75% 아미노산 서열 동일성을 갖는 아미노산 서열을 포함하는, 방법.
- 청구항 64에 있어서, 상기 효소 활성은 표적 DNA를 변형시키는, 방법.
- 청구항 74에 있어서, 상기 효소 활성은 뉴클레아제 활성, 메틸전이효소 활성, 탈메틸효소 활성, DNA 치유 활성, DNA 손상 활성, 탈아미노화 활성, 디스무타제 활성, 알킬화 활성, 탈퓨린화 활성, 산화 활성, 피리미딘 이량체 형성 활성, 인테그라제 활성, 전위효소 활성, 재조합효소 활성, 폴리머라제 활성, 리가제 활성, 헬리카제 활성, 광분해효소 활성 또는 글리코실라제 활성인, 방법.
- 청구항 75에 있어서, 상기 DNA-변형 효소 활성은 뉴클레아제 활성인, 방법.
- 청구항 76에 있어서, 상기 뉴클레아제 활성은 표적 DNA에서 이중 가닥 절단을 도입하는, 방법.
- 청구항 77에 있어서, 상기 접촉은 비상동 말단 연결 또는 상동성-유도된 치유에 대해 허용된 조건 하에서 일어나는, 방법.
- 청구항 78에 있어서, 표적 DNA를 공여체 폴리뉴클레오타이드와 접촉시키는 것을 추가로 포함하고, 상기 공여체 폴리뉴클레오타이드, 공여체 폴리뉴클레오타이드의 부분, 공여체 폴리뉴클레오타이드의 카피, 또는 공여체 폴리뉴클레오타이드의 카피의 부분은 표적 DNA에 통합되는, 방법.
- 청구항 78에 있어서, 상기 방법은 세포를 공여체 폴리뉴클레오타이드와 접촉시키는 것을 포함하지 않고, 상기 표적 DNA는 변형되고 이로써 상기 표적 DNA 내의 뉴클레오타이드는 결실되는, 방법.
- 청구항 64에 있어서, 상기 효소 활성은 표적 DNA와 연관된 표적 폴리펩타이드를 변형시키는, 방법.
- 청구항 81에 있어서, 상기 효소 활성은 메틸전이효소 활성, 탈메틸효소 활성, 아세틸전이효소 활성, 탈아세틸화효소 활성, 키나아제 활성, 포스파타제 활성, 우비퀴틴 리가제 활성, 탈우비퀴틴화 활성, 아데닐화 활성, 탈아데닐화 활성, 스모화 활성, 탈스모화 활성, 리보실화 활성, 탈리보실화 활성, 미리스토일화 활성 또는 탈미리스토일화 활성인, 방법.
- 청구항 81에 있어서, 상기 표적 폴리펩타이드는 히스톤이고 상기 효소 활성은 메틸전이효소 활성, 탈메틸효소 활성, 아세틸전이효소 활성, 탈아세틸화효소 활성, 키나아제 활성, 포스파타제 활성, 우비퀴틴 리가제 활성 또는 탈우비퀴틴화 활성인, 방법.
- 청구항 64에 있어서, 상기 복합물은 활성제-RNA를 추가로 포함하는, 방법.
- 청구항 84에 있어서, 상기 활성제-RNA는 적어도 8 개의 인접 뉴클레오타이드의 스트레치에 대해 서열번호:431-682에서 제시된 뉴클레오타이드 서열의 임의의 하나와 적어도 60% 동일성을 갖는 뉴클레오타이드 서열을 포함하는, 방법.
- 표적 DNA를 하기와 접촉시키는 단계를 포함하는, 표적 DNA 내에서 부위 특이적인 전사를 조절하는 방법으로서:
(i) 하기를 포함하는 DNA-표적화 RNA, 또는 이것을 인코딩하는 DNA 폴리뉴클레오타이드,:
(a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및
(b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절; 및
(ii) 하기를 포함하는 부위 지향적 변형 폴리펩타이드, 또는 그것을 인코딩하는 폴리뉴클레오타이드:
(a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및
(b) 전사를 조절하는 활성부,
상기 접촉은 표적 DNA 내에서 전사의 조절을 야기하는, 방법. - 청구항 86에 있어서, 상기 표적 DNA 내의 전사는 증가된 방법.
- 청구항 86에 있어서, 상기 표적 DNA 내의 전사는 감소된 방법.
- 표적 DNA를 하기와 접촉시키는 단계를 포함하는, 표적 DNA에서 부위 특이적인 변형의 방법:
(i) 하기를 포함하는 DNA-표적화 RNA, 또는 이것을 인코딩하는 DNA 폴리뉴클레오타이드:
(a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및
(b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절; 및
(ii) 하기를 포함하는 부위 지향적 변형 폴리펩타이드, 또는 그것을 인코딩하는 폴리뉴클레오타이드:
(a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및
(b) 표적 DNA 내에서 전사를 조절하는 활성부. - 청구항 86에 있어서, 상기 부위 지향적 변형 폴리펩타이드는 표적 DNA 내에서 전사를 증가시키는, 방법.
- 청구항 86에 있어서, 상기 부위 지향적 변형 폴리펩타이드는 표적 DNA 내에서 전사를 감소시키는, 방법.
- 세포에 하기를 도입하는 것을 포함하는, 세포에서 표적 DNA의 부위 특이적인 절단 및 변형을 촉진하는 방법으로서:
(i) 하기를 포함하는 DNA-표적화 RNA, 또는 이것을 인코딩하는 DNA 폴리뉴클레오타이드:
(a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및
(b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절; 및
(ii) 하기를 포함하는 부위 지향적 변형 폴리펩타이드, 또는 그것을 인코딩하는 폴리뉴클레오타이드:
(a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및
(b) 표적 DNA에서 이중 가닥 절단을 창출하는 뉴클레아제 활성을 나타내는 활성부;
상기 이중 가닥 절단의 부위는 DNA-표적화 RNA에 의해 결정되고, 상기 접촉은 비상동 말단 연결 또는 상동성-유도된 치유에 대해 허용된 조건 하에서 일어나고, 상기 표적 DNA는 절단되고 재연결되어 변형된 DNA 서열을 생산하는, 방법. - 청구항 92에 있어서, 표적 DNA를 공여체 폴리뉴클레오타이드와 접촉시키는 것을 추가로 포함하고, 상기 공여체 폴리뉴클레오타이드, 공여체 폴리뉴클레오타이드의 부분, 공여체 폴리뉴클레오타이드의 카피, 또는 공여체 폴리뉴클레오타이드의 카피의 부분은 표적 DNA에 통합되는, 방법.
- 청구항 92에 있어서, 상기 방법은 세포를 공여체 폴리뉴클레오타이드와 접촉시키는 것을 포함하지 않고, 상기 표적 DNA는 변형되고 이로써 상기 표적 DNA 내의 뉴클레오타이드는 결실되는, 방법.
- 청구항 92에 있어서, 상기 세포는 하기로 이루어진 그룹으로부터 선택되는 방법: 고세균 세포, 박테리아 세포, 진핵 세포, 진핵 단일-세포 유기체, 체세포, 생식세포, 줄기세포, 식물 세포, 조류 세포, 동물 세포, 무척추동물 세포, 척추동물 세포, 물고기 세포, 개구리 세포, 새 세포, 포유동물 세포, 돼지 세포, 암소 세포, 염소 세포, 양 세포, 설치류 세포, 랫트 세포, 마우스 세포, 비-인간 영장류 세포, 및 인간 세포.
- 청구항 92에 있어서, 상기 세포는 시험관 내에 있는, 방법.
- 청구항 92에 있어서, 상기 세포는 생체 내에 있는, 방법.
- 하기를 포함하는, 대상체에서 유전적으로 변형된 세포를 생산하는 방법:
(I) 세포에 하기를 도입하는 단계로서:
(i) 하기를 포함하는 DNA-표적화 RNA, 또는 이것을 인코딩하는 DNA 폴리뉴클레오타이드:
(a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및
(b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절; 및
(ii) 하기를 포함하는 부위 지향적 변형 폴리펩타이드, 또는 그것을 인코딩하는 폴리뉴클레오타이드:
(a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및
(b) 표적 DNA에서 이중 가닥 절단을 창출하는 뉴클레아제 활성을 나타내는 활성부;
상기 이중 가닥 절단의 부위는 DNA-표적화 RNA에 의해 결정되고, 상기 접촉은 비상동 말단 연결 또는 상동성-유도된 치유에 대해 허용된 조건 하에서 일어나고, 상기 표적 DNA는 절단되고 재연결되어 변형된 DNA 서열을 생산하고;
그렇게 함으로써 유전적으로 변형된 세포를 생산하는 단계; 및
(II) 상기 유전적으로 변형된 세포를 상기 대상체에 이식하는 단계. - 청구항 98에 있어서, 세포를 공여체 폴리뉴클레오타이드와 접촉시키는 것을 추가로 포함하는, 여기서 상기 공여체 폴리뉴클레오타이드, 공여체 폴리뉴클레오타이드의 부분, 공여체 폴리뉴클레오타이드의 카피, 또는 공여체 폴리뉴클레오타이드의 카피의 부분은 표적 DNA에 통합되는, 방법.
- 청구항 98에 있어서, 상기 방법은 세포를 공여체 폴리뉴클레오타이드와 접촉시키는 것을 포함하지 않고, 상기 표적 DNA는 변형되고 이로써 상기 표적 DNA 내의 뉴클레오타이드는 결실되는, 방법.
- 청구항 98에 있어서, 상기 세포는 하기로 이루어진 그룹으로부터 선택되는 방법: 고세균 세포, 박테리아 세포, 진핵 세포, 진핵 단일-세포 유기체, 체세포, 생식세포, 줄기세포, 식물 세포, 조류 세포, 동물 세포, 무척추동물 세포, 척추동물 세포, 물고기 세포, 양서류 세포, 새 세포, 포유동물 세포, 유제류 세포, 설치류 세포, 비-인간 영장류 세포, 및 인간 세포.
- 외인성 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 유전적으로 변형된 세포에서 표적 DNA를 조절하는 방법으로서, 상기 방법은 유전적으로 변형된 세포에 DNA-표적화 RNA, 또는 이것을 인코딩하는 DNA 폴리뉴클레오타이드를 도입하는 것을 포함하고, 여기서:
(i) 상기 DNA-표적화 RNA는 하기:
(a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및
(b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절을 포함하고;
(ii) 상기 부위 지향적 변형 폴리펩타이드는 하기:
(a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및
(b) 뉴클레아제 활성을 나타내는 활성부를 포함하는, 방법. - 청구항 102에 있어서, 상기 부위 지향적 변형 폴리펩타이드는 도 3에서 묘사된 Cas9/Csn1 아미노산 서열의 아미노산 7-166 또는 731-1003과, 또는 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 상응하는 부분과 적어도 약 75% 아미노산 서열 동일성을 갖는 아미노산 서열을 포함하는, 방법.
- 청구항 102에 있어서, 상기 세포는 하기로 이루어진 그룹으로부터 선택되는 방법: 고세균 세포, 박테리아 세포, 진핵 세포, 진핵 단일-세포 유기체, 체세포, 생식세포, 줄기세포, 식물 세포, 조류 세포, 동물 세포, 무척추동물 세포, 척추동물 세포, 물고기 세포, 양서류 세포, 새 세포, 포유동물 세포, 유제류 세포, 설치류 세포, 비-인간 영장류 세포, 및 인간 세포.
- 청구항 102에 있어서, 상기 세포는 생체 내에 있는, 방법.
- 청구항 102에 있어서, 상기 세포는 시험관 내에 있는, 방법.
- 청구항 102에 있어서, 상기 부위 지향적 변형 폴리펩타이드의 발현은 유도성 프로모터의 조절 하에 있는, 방법.
- 청구항 102에 있어서, 상기 부위 지향적 변형 폴리펩타이드의 발현은 세포형-특이적 프로모터의 조절 하에 있는, 방법.
- 하기를 포함하는 키트:
(i) 청구항 1의 DNA-표적화 RNA, 또는 이것을 인코딩하는 DNA 폴리뉴클레오타이드; 및
(ii) 재구성 및/또는 희석을 위한 시약. - 청구항 109에 있어서, 하기로 이루어진 그룹으로부터 선택된 시약을 추가로 포함하는 키트: 세포에 DNA-표적화 RNA를 도입하기 위한 완충제, 세척 완충제, 대조군 시약, 대조군 발현 벡터 또는 RNA 폴리뉴클레오타이드, DNA로부터 DNA-표적화 RNA를 전사시키기 위한 시약, 및 이들의 조합.
- 하기를 포함하는 키트:
(i) 청구항 44의 부위 지향적 변형 폴리펩타이드, 또는 그것을 인코딩하는 폴리뉴클레오타이드; 및
(ii) 재구성 및/또는 희석을 위한 시약. - 청구항 111에 있어서, 하기로 이루어진 그룹으로부터 선택된 시약을 추가로 포함하는 키트: 세포에 부위 지향적 변형 폴리펩타이드를 도입하기 위한 완충제, 세척 완충제, 대조군 시약, 대조군 발현 벡터 또는 RNA 폴리뉴클레오타이드, DNA로부터 부위 지향적 변형 폴리펩타이드의 시험관내 생산을 위한 시약, 및 이들의 조합.
- 하기를 포함하는 키트:
(i) 청구항 60의 부위 지향적 변형 폴리펩타이드, 또는 그것을 인코딩하는 폴리뉴클레오타이드; 및
(ii) 재구성 및/또는 희석을 위한 시약 - 하기를 포함하는 키트:
(i) 하기를 포함하는 DNA-표적화 RNA, 또는 이것을 인코딩하는 DNA 폴리뉴클레오타이드:
(a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및
(b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절; 및
(ii) 하기를 포함하는 부위 지향적 변형 폴리펩타이드, 또는 그것을 인코딩하는 폴리뉴클레오타이드:
(a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및
(b) 부위 지향적 효소 활성을 나타내는 활성부로서, 상기 효소 활성의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부. - 하기를 포함하는 키트:
(i) 하기를 포함하는 DNA-표적화 RNA, 또는 이것을 인코딩하는 DNA 폴리뉴클레오타이드:
(a) 표적 DNA에서 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절; 및
(b) 부위 지향적 변형 폴리펩타이드와 상호작용하는 제 2 분절; 및
(ii) 하기를 포함하는 상기 부위 지향적 변형 폴리펩타이드, 또는 그것을 인코딩하는 폴리뉴클레오타이드:
(a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및
(b) 표적 DNA 내에서 전사를 조절하는 활성부로서, 상기 표적 DNA 내의 조절된 전사의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부. - 하기를 포함하는 키트:
(i) 청구항 12의 재조합 발현 벡터; 및
(ii) 재구성 및/또는 희석을 위한 시약 - 하기를 포함하는 키트:
(i) 청구항 13의 재조합 발현 벡터; 및
(ii) 재구성 및/또는 희석을 위한 시약 - 하기를 포함하는 키트:
(i) 청구항 7의 재조합 발현 벡터; 및
(ii) 재구성 및/또는 희석을 위한 시약 - 하기를 포함하는 키트:
(i) 청구항 7의 재조합 발현 벡터; 및
(ii) 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 재조합 발현 벡터로서, 상기 부위 지향적 변형 폴리펩타이드는 하기를 포함하는 재조합 발현 벡터:
(a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및
(b) 부위 지향적 효소 활성을 나타내는 활성부로서, 상기 효소 활성의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부. - 하기를 포함하는 키트:
(i) 청구항 7의 재조합 발현 벡터; 및
(ii) 부위 지향적 변형 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 재조합 발현 벡터로서, 상기 부위 지향적 변형 폴리펩타이드는 하기를 포함하는 재조합 발현 벡터:
(a) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및
(b) 표적 DNA 내에서 전사를 조절하는 활성부로서, 상기 표적 DNA 내의 조절된 전사의 부위는 DNA-표적화 RNA에 의해 결정되는 활성부. - 청구항 1에 따른 2 이상의 DNA-표적화 RNA, 또는 그것을 인코딩하는 DNA 폴리뉴클레오타이드를 포함하는 표적 DNA를 표적으로 하기 위한 키트로서, 상기 2 이상의 DNA-표적화 RNA의 적어도 하나의 제 1 분절은 2 이상의 DNA-표적화 RNA의 적어도 하나의 제 1 분절의 다른 것으로부터 적어도 하나의 뉴클레오타이드와 다른 키트.
- 숙주세포에서 표적 DNA의 전사를 선택적으로 조절하는 방법으로서, 상기 방법은 숙주세포에 하기를 도입하는 것을 포함하고:
a) 하기를 포함하는 DNA-표적화 RNA, 또는 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산:
i) 표적 DNA에서 표적 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절;
ii) 부위 지향적 폴리펩타이드와 상호작용하는 제 2 분절; 및
iii) 안정성 대조군 서열; 및
b) 하기를 포함하는 변이체 Cas9 부위 지향적 폴리펩타이드, 또는 변이체 Cas9 부위 지향적 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산:
i) DNA-표적화 RNA와 상호작용하는 RNA-결합부; 및
ii) 감소된 엔도데옥시리보뉴클레아제 활성을 나타내는 활성부,
상기 DNA-표적화 RNA 및 상기 변이체 Cas9 폴리펩타이드는 세포에서 복합물을 형성하고 여기서 상기 복합물은 세포에서 표적 DNA의 전사를 선택적으로 조절하는, 방법. - 청구항 122에 있어서, 상기 변이체 Cas9 부위 지향적 폴리펩타이드는 비-표적 DNA의 상보적 가닥을 절단할 수 있지만 표적 DNA의 상보적 가닥을 절단하기 위한 감소된 능력을 갖는, 방법.
- 청구항 123에 있어서, 상기 변이체 부위 지향적 폴리펩타이드는 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 도 39a에서 묘사된 아미노산 서열의 H840A 돌연변이 또는 상응하는 돌연변이를 포함하는, 방법.
- 청구항 122에 있어서, 상기 변이체 부위 지향적 폴리펩타이드는 표적 DNA의 상보적 가닥을 절단할 수 있지만 비-표적 DNA의 상보적 가닥을 절단하기 위한 감소된 능력을 갖는, 방법.
- 청구항 125에 있어서, 상기 변이체 부위 지향적 폴리펩타이드는 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 도 39a에서 묘사된 아미노산 서열의 D10A 돌연변이 또는 상응하는 돌연변이를 포함하는, 방법.
- 청구항 122에 있어서, 상기 변이체 부위 지향적 폴리펩타이드는 표적 DNA의 상보적 가닥을 절단하기 위한 감소된 능력 및 비-표적 DNA의 상보적 가닥을 절단하기 위한 감소된 능력을 갖는, 방법.
- 청구항 127에 있어서, 상기 변이체 부위 지향적 폴리펩타이드는 하기의 둘 모두를 포함하는 방법: (i) 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 도 41에서 묘사된 아미노산 서열의 D10A 돌연변이 또는 상응하는 돌연변이; 및 (ii) 서열번호:1-256 및 795-1346에서 제시된 아미노산 서열의 어떤 것에서 서열번호:8의 H840A 돌연변이 또는 상응하는 돌연변이.
- 청구항 122에 있어서, 상기 숙주세포는 원핵 세포인, 방법.
- 청구항 122에 있어서, 상기 숙주세포는 진핵 세포인, 방법.
- 청구항 130에 있어서, 상기 진핵 세포는 포유동물 세포인, 방법.
- 청구항 122에 있어서 상기 표적 DNA의 전사는 복합물의 부재에서 표적 DNA의 전사와 비교하여 적어도 약 10%까지 억제되는, 방법.
- 청구항 122에 있어서, 상기 변이체 Cas9 부위 지향적 폴리펩타이드는 이종기원 폴리펩타이드를 포함하는, 방법.
- 청구항 133에 있어서, 상기 이종기원 폴리펩타이드는 준세포 국재화 서열, 검출가능한 표지, 안정성 대조군 펩타이드, 전사 조절 서열, 단백질 결합 서열, 또는 이들의 조합을 포함하는, 방법.
- 청구항 134에 있어서, 상기 이종기원 폴리펩타이드는 안정성 대조군 펩타이드를 포함하는, 방법.
- 청구항 135에 있어서, 상기 안정성 대조군 펩타이드는 데그론 서열을 포함하는, 방법.
- 청구항 133에 있어서, 상기 이종기원 폴리펩타이드는 하기로 이루어진 그룹으로부터 선택되는 방법: 전사 활성제, 전사 억제인자, 히스톤 라이신 메틸전이효소, 히스톤 라이신 탈메틸효소, 히스톤 라이신 아세틸전이효소, 히스톤 라이신 탈아세틸화효소, DNA 메틸화효소, DNA 탈메틸효소, 경계 요소, 주변 동원 요소, 단백질 접합 요소, 및 이들의 조합.
- 청구항 133에 있어서, 상기 표적 DNA의 전사는 복합물의 부재에서 표적 DNA의 전사와 비교하여 적어도 약 1.2 배까지 증가되는, 방법.
- 청구항 138에 있어서, 상기 이종기원 폴리펩타이드는 하기로 이루어진 그룹으로부터 선택되는 방법: 전사 활성제, 히스톤 라이신 메틸전이효소, 히스톤 라이신 탈메틸효소, 히스톤 라이신 아세틸전이효소, DNA 탈메틸효소, 단백질 접합 요소, 및 이들의 조합.
- 청구항 122에 있어서, 상기 DNA-표적화 RNA는 단일-분자 DNA-표적화 RNA인, 방법.
- 청구항 122에 있어서, 상기 DNA-표적화 RNA는 2-분자 DNA-표적화 RNA인, 방법.
- 청구항 122에 있어서, 숙주세포에 제 2 DNA-표적화 RNA, 또는 제 2 DNA-표적화 RNA을 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산을 도입하는 것을 추가로 포함하고, 여기서 제 2 DNA-표적화 RNA는 표적 DNA에서 제 2 표적 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절;
부위 지향적 폴리펩타이드와 상호작용하는 제 2 분절; 및
안정성 대조군 서열을 포함하는, 방법. - 청구항 122에 있어서, 상기 제 2 분절은 약 30 개의 뉴클레오타이드 내지 약 60 개의 뉴클레오타이드의 길이를 가지며, 하기로부터 선택된 뉴클레오타이드 서열에 대해 적어도 약 95% 뉴클레오타이드 서열 동일성을 갖는 뉴클레오타이드 서열을 포함하는 방법:
1) 5'-GUUUUAGAGCUA-(링커)-UAGCAAGUUAAAA-3';
2) 5'-GUUUAGAGCUG-(링커)-CAGCAAGUUAAA-3';
3) 5'-GUUUUAGAGCUG-(링커)-CAGCGAGUUAAA-3';
4) 5'-GUUUUUGUACUCU-(링커)-AGAAGCUACAAAGAU-3';
5) 5'-GUUGUAGCUCC-(링커)-GUUGCUACAAU-3'; 및
6) 5'-GUUUUAGAGCUAGAAAUAGCAAGUUAAAAUAAGGCUAGUCCG-3'. - 하기를 포함하는 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 단리된 핵산:
a) 표적 DNA에서 표적 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절;
b) 부위 지향적 폴리펩타이드와 상호작용하는 제 2 분절; 및
c) 안정성 대조군 서열. - 청구항 144에 있어서, 상기 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열은 프로모터에 작동가능하게 연결되는, 단리된 핵산.
- 청구항 144에 있어서, 상기 핵산은 야생형 Cas9에 대해 감소된 엔도데옥시리보뉴클레아제 활성을 나타내는 변이체 Cas9 부위 지향적 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 추가로 포함하는, 단리된 핵산.
- 청구항 146에 있어서, 상기 변이체 Cas9 부위 지향적 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열은 프로모터에 작동가능하게 연결되는, 단리된 핵산.
- 청구항 24의 핵산을 포함하는, 재조합 발현 벡터.
- 청구항 24의 단리된 핵산, 또는 청구항 28의 재조합 발현 벡터를 포함하는, 유전적으로 변형된 숙주세포.
- 복수의 멤버를 포함하는 핵산의 라이브러리로서, 각 멤버 핵산은,
a) 표적 DNA에서 표적 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절;
b) 부위 지향적 폴리펩타이드와 상호작용하는 제 2 분절; 및
c) 안정성 대조군 서열을 포함하는 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하고,
각 멤버는 제 1 분절의 뉴클레오타이드 서열에서 라이브러리의 다른 멤버와 상이한 라이브러리. - 청구항 150에 있어서, 상기 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열은 프로모터에 작동가능하게 연결되는, 라이브러리.
- 하기를 포함하는 키트:
a) 하기를 포함하는 DNA-표적화 RNA, 또는 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산:
i) 표적 DNA에서 표적 서열에 대해 상보적인 뉴클레오타이드 서열을 포함하는 제 1 분절;
ii) 부위 지향적 폴리펩타이드와 상호작용하는 제 2 분절; 및
iii) 안정성 대조군 서열; 및
b) 완충제. - 청구항 152에 있어서, 야생형 Cas9에 대해 감소된 엔도데옥시리보뉴클레아제 활성을 나타내는 변이체 Cas9 부위 지향적 폴리펩타이드를 추가로 포함하는 키트.
- 청구항 152에 있어서, 상기 DNA-표적화 RNA를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산은 야생형 Cas9에 대해 감소된 엔도데옥시리보뉴클레아제 활성을 나타내는 변이체 Cas9 부위 지향적 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 추가로 포함하는 키트.
- 청구항 152에 있어서, 야생형 Cas9에 대해 감소된 엔도데옥시리보뉴클레아제 활성을 나타내는 변이체 Cas9 부위 지향적 폴리펩타이드를 인코딩하는 뉴클레오타이드 서열을 포함하는 핵산을 추가로 포함하는 키트.
Applications Claiming Priority (9)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201261652086P | 2012-05-25 | 2012-05-25 | |
| US61/652,086 | 2012-05-25 | ||
| US201261716256P | 2012-10-19 | 2012-10-19 | |
| US61/716,256 | 2012-10-19 | ||
| US201361757640P | 2013-01-28 | 2013-01-28 | |
| US61/757,640 | 2013-01-28 | ||
| US201361765576P | 2013-02-15 | 2013-02-15 | |
| US61/765,576 | 2013-02-15 | ||
| PCT/US2013/032589 WO2013176772A1 (en) | 2012-05-25 | 2013-03-15 | Methods and compositions for rna-directed target dna modification and for rna-directed modulation of transcription |
Related Child Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020177034069A Division KR20170134766A (ko) | 2012-05-25 | 2013-03-15 | Rna-유도된 표적 dna 변형 및 전사의 rna-유도된 조절을 위한 방법 및 조성물 |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20150016588A true KR20150016588A (ko) | 2015-02-12 |
Family
ID=49624232
Family Applications (2)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020177034069A Ceased KR20170134766A (ko) | 2012-05-25 | 2013-03-15 | Rna-유도된 표적 dna 변형 및 전사의 rna-유도된 조절을 위한 방법 및 조성물 |
| KR1020147036096A Ceased KR20150016588A (ko) | 2012-05-25 | 2013-03-15 | Rna-유도된 표적 dna 변형 및 전사의 rna-유도된 조절을 위한 방법 및 조성물 |
Family Applications Before (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020177034069A Ceased KR20170134766A (ko) | 2012-05-25 | 2013-03-15 | Rna-유도된 표적 dna 변형 및 전사의 rna-유도된 조절을 위한 방법 및 조성물 |
Country Status (41)
Cited By (16)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| KR20150056539A (ko) * | 2012-07-25 | 2015-05-26 | 더 브로드 인스티튜트, 인코퍼레이티드 | 유도 dna 결합 단백질 및 게놈 교란 도구 및 이의 적용 |
| KR20150101476A (ko) * | 2012-10-23 | 2015-09-03 | 주식회사 툴젠 | 표적 DNA에 특이적인 가이드 RNA 및 Cas 단백질을 암호화하는 핵산 또는 Cas 단백질을 포함하는, 표적 DNA를 절단하기 위한 조성물 및 이의 용도 |
| KR20170124565A (ko) * | 2015-03-03 | 2017-11-10 | 더 제너럴 하스피탈 코포레이션 | 변경된 PAM 특이성을 갖는 조작된 CRISPR-Cas9 뉴클레아제 |
| KR20180085795A (ko) * | 2015-12-04 | 2018-07-27 | 카리부 바이오사이언시스 인코포레이티드 | 조작된 핵산 표적화 핵산 |
| WO2018208067A1 (ko) * | 2017-05-08 | 2018-11-15 | 주식회사 툴젠 | 인위적으로 조작된 조작면역세포 |
| KR20190046071A (ko) | 2017-10-25 | 2019-05-07 | 성균관대학교산학협력단 | 노화 모델 제조 방법 및 이에 의해 제조된 세포 또는 동물 노화 모델 |
| KR20200016892A (ko) * | 2017-06-09 | 2020-02-17 | 에디타스 메디신, 인코포레이티드 | 조작된 cas9 뉴클레아제 |
| KR20200123255A (ko) * | 2018-03-26 | 2020-10-28 | 고쿠리츠다이가쿠호진 고베다이가쿠 | 세포에서 이중 가닥 dna의 표적 부위를 변형시키기 위한 방법 |
| KR20210013288A (ko) * | 2012-10-23 | 2021-02-03 | 주식회사 툴젠 | 표적 DNA에 특이적인 가이드 RNA 및 Cas 단백질을 암호화하는 핵산 또는 Cas 단백질을 포함하는, 표적 DNA를 절단하기 위한 조성물 및 이의 용도 |
| KR20230022743A (ko) | 2021-08-09 | 2023-02-16 | 경상국립대학교산학협력단 | 콩 유전자교정 효율 증대를 위한 유전자교정 시스템 및 이의 용도 |
| KR20230022745A (ko) | 2021-08-09 | 2023-02-16 | 경상국립대학교산학협력단 | P34와 이의 상동체 동시 타겟 유전자교정 시스템 및 이의 용도 |
| KR20230022747A (ko) | 2021-08-09 | 2023-02-16 | 경상국립대학교산학협력단 | Mips1과 이의 상동체 동시 타겟 유전자교정 시스템 및 이의 용도 |
| KR20230022748A (ko) | 2021-08-09 | 2023-02-16 | 경상국립대학교산학협력단 | E2와 이의 상동체 동시 타겟 유전자교정 시스템 및 이의 용도 |
| KR20230022749A (ko) | 2021-08-09 | 2023-02-16 | 경상국립대학교산학협력단 | GmIPK1 유전자교정 시스템 및 이의 용도 |
| KR20230051095A (ko) | 2021-10-06 | 2023-04-17 | 주식회사 진코어 | 유전자 편집을 위한 TaRGET 시스템 및 이의 용도 |
| US12012598B2 (en) | 2016-08-12 | 2024-06-18 | Toolgen Incorporated | Manipulated immunoregulatory element and immunity altered thereby |
Families Citing this family (1475)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| EP2401367B1 (en) | 2009-02-26 | 2016-11-30 | Transposagen Biopharmaceuticals, Inc. | Hyperactive piggybac transposases |
| ES2978496T3 (es) | 2009-07-08 | 2024-09-13 | Kymab Ltd | Modelos animales y moléculas terapéuticas |
| US9445581B2 (en) | 2012-03-28 | 2016-09-20 | Kymab Limited | Animal models and therapeutic molecules |
| US9585920B2 (en) | 2011-02-04 | 2017-03-07 | Katherine Rose Kovarik | Method and system for treating cancer cachexia |
| US9457077B2 (en) | 2009-11-18 | 2016-10-04 | Katherine Rose Kovarik | Method and system for targeting the microbiome to promote health and treat allergic and inflammatory diseases |
| US9719068B2 (en) | 2010-05-06 | 2017-08-01 | Children's Hospital Medical Center | Methods and systems for converting precursor cells into intestinal tissues through directed differentiation |
| US10548761B2 (en) | 2011-02-04 | 2020-02-04 | Joseph E. Kovarik | Method and system for reducing the likelihood of colorectal cancer in a human being |
| US10512661B2 (en) | 2011-02-04 | 2019-12-24 | Joseph E. Kovarik | Method and system for reducing the likelihood of developing liver cancer in an individual diagnosed with non-alcoholic fatty liver disease |
| US10086018B2 (en) | 2011-02-04 | 2018-10-02 | Joseph E. Kovarik | Method and system for reducing the likelihood of colorectal cancer in a human being |
| US10687975B2 (en) | 2011-02-04 | 2020-06-23 | Joseph E. Kovarik | Method and system to facilitate the growth of desired bacteria in a human's mouth |
| US10940169B2 (en) | 2015-11-30 | 2021-03-09 | Joseph E. Kovarik | Method for reducing the likelihood of developing cancer in an individual human being |
| US11844720B2 (en) | 2011-02-04 | 2023-12-19 | Seed Health, Inc. | Method and system to reduce the likelihood of dental caries and halitosis |
| US10842834B2 (en) | 2016-01-06 | 2020-11-24 | Joseph E. Kovarik | Method and system for reducing the likelihood of developing liver cancer in an individual diagnosed with non-alcoholic fatty liver disease |
| US11273187B2 (en) | 2015-11-30 | 2022-03-15 | Joseph E. Kovarik | Method and system for reducing the likelihood of developing depression in an individual |
| US11951140B2 (en) | 2011-02-04 | 2024-04-09 | Seed Health, Inc. | Modulation of an individual's gut microbiome to address osteoporosis and bone disease |
| US10835560B2 (en) | 2013-12-20 | 2020-11-17 | Joseph E. Kovarik | Reducing the likelihood of skin cancer in an individual human being |
| US11191665B2 (en) | 2011-02-04 | 2021-12-07 | Joseph E. Kovarik | Method and system for reducing the likelihood of a porphyromonas gingivalis infection in a human being |
| US11523934B2 (en) | 2011-02-04 | 2022-12-13 | Seed Health, Inc. | Method and system to facilitate the growth of desired bacteria in a human's mouth |
| US10583033B2 (en) | 2011-02-04 | 2020-03-10 | Katherine Rose Kovarik | Method and system for reducing the likelihood of a porphyromonas gingivalis infection in a human being |
| US11951139B2 (en) | 2015-11-30 | 2024-04-09 | Seed Health, Inc. | Method and system for reducing the likelihood of osteoporosis |
| US11357722B2 (en) | 2011-02-04 | 2022-06-14 | Seed Health, Inc. | Method and system for preventing sore throat in humans |
| US12279989B2 (en) | 2011-02-04 | 2025-04-22 | Seed Health, Inc. | Method and system for increasing beneficial bacteria and decreasing pathogenic bacteria in the oral cavity |
| US9987224B2 (en) | 2011-02-04 | 2018-06-05 | Joseph E. Kovarik | Method and system for preventing migraine headaches, cluster headaches and dizziness |
| US11419903B2 (en) | 2015-11-30 | 2022-08-23 | Seed Health, Inc. | Method and system for reducing the likelihood of osteoporosis |
| US10314865B2 (en) | 2011-02-04 | 2019-06-11 | Katherine Rose Kovarik | Method and system for treating cancer and other age-related diseases by extending the healthspan of a human |
| US10085938B2 (en) | 2011-02-04 | 2018-10-02 | Joseph E. Kovarik | Method and system for preventing sore throat in humans |
| US10010568B2 (en) | 2011-02-04 | 2018-07-03 | Katherine Rose Kovarik | Method and system for reducing the likelihood of a spirochetes infection in a human being |
| US10245288B2 (en) | 2011-02-04 | 2019-04-02 | Joseph E. Kovarik | Method and system for reducing the likelihood of developing NASH in an individual diagnosed with non-alcoholic fatty liver disease |
| US12257272B2 (en) | 2015-12-24 | 2025-03-25 | Seed Health, Inc. | Method and system for reducing the likelihood of developing depression in an individual |
| US11998479B2 (en) | 2011-02-04 | 2024-06-04 | Seed Health, Inc. | Method and system for addressing adverse effects on the oral microbiome and restoring gingival health caused by sodium lauryl sulphate exposure |
| US9730967B2 (en) | 2011-02-04 | 2017-08-15 | Katherine Rose Kovarik | Method and system for treating cancer cachexia |
| US10111913B2 (en) | 2011-02-04 | 2018-10-30 | Joseph E. Kovarik | Method of reducing the likelihood of skin cancer in an individual human being |
| AR085533A1 (es) | 2011-03-23 | 2013-10-09 | Pioneer Hi Bred Int | Metodos de produccion de un locus de rasgos transgenicos complejo |
| KR102025295B1 (ko) | 2011-06-30 | 2019-09-26 | 애로우헤드 파마슈티컬스 인코포레이티드 | B형 간염 바이러스의 유전자 발현 저해용 조성물 및 방법 |
| CA2853829C (en) | 2011-07-22 | 2023-09-26 | President And Fellows Of Harvard College | Evaluation and improvement of nuclease cleavage specificity |
| JP5813880B2 (ja) | 2011-09-19 | 2015-11-17 | カイマブ・リミテッド | ヒトへの使用に合わせて作製された抗体、可変ドメインおよび鎖 |
| WO2013045916A1 (en) | 2011-09-26 | 2013-04-04 | Kymab Limited | Chimaeric surrogate light chains (slc) comprising human vpreb |
| US10465042B2 (en) | 2011-12-02 | 2019-11-05 | Yale University | Poly(amine-co-ester) nanoparticles and methods of use thereof |
| US9253965B2 (en) | 2012-03-28 | 2016-02-09 | Kymab Limited | Animal models and therapeutic molecules |
| US11458157B2 (en) | 2011-12-16 | 2022-10-04 | Targetgene Biotechnologies Ltd. | Compositions and methods for modifying a predetermined target nucleic acid sequence |
| WO2014163886A1 (en) | 2013-03-12 | 2014-10-09 | President And Fellows Of Harvard College | Method of generating a three-dimensional nucleic acid containing matrix |
| ES2991004T3 (es) | 2011-12-22 | 2024-12-02 | Harvard College | Métodos para la detección de analitos |
| GB201122458D0 (en) | 2011-12-30 | 2012-02-08 | Univ Wageningen | Modified cascade ribonucleoproteins and uses thereof |
| US9637739B2 (en) | 2012-03-20 | 2017-05-02 | Vilnius University | RNA-directed DNA cleavage by the Cas9-crRNA complex |
| GB2502127A (en) | 2012-05-17 | 2013-11-20 | Kymab Ltd | Multivalent antibodies and in vivo methods for their production |
| US10251377B2 (en) | 2012-03-28 | 2019-04-09 | Kymab Limited | Transgenic non-human vertebrate for the expression of class-switched, fully human, antibodies |
| EP2847335B1 (en) | 2012-04-25 | 2018-06-27 | Regeneron Pharmaceuticals, Inc. | Nuclease-mediated targeting with large targeting vectors |
| EP3597741A1 (en) | 2012-04-27 | 2020-01-22 | Duke University | Genetic correction of mutated genes |
| LT2800811T (lt) | 2012-05-25 | 2017-09-11 | The Regents Of The University Of California | Būdai ir kompozicijos, skirti tikslinės dnr modifikavimui, panaudojant adresuotą rnr, ir transkripcijos moduliavimui, panaudojant adresuotą rnr |
| WO2013177560A1 (en) | 2012-05-25 | 2013-11-28 | The Regents Of The University Of California | Microfluidic systems for particle trapping and separation |
| WO2013188522A2 (en) * | 2012-06-12 | 2013-12-19 | Genentech, Inc. | Methods and compositions for generating conditional knock-out alleles |
| WO2013192278A1 (en) | 2012-06-19 | 2013-12-27 | Regents Of The University Of Minnesota | Gene targeting in plants using dna viruses |
| WO2014078819A2 (en) | 2012-11-16 | 2014-05-22 | Transposagen Biopharmaceuticals, Inc. | Site-specific enzymes and methods of use |
| AU2013352156B2 (en) | 2012-11-27 | 2018-12-06 | Children's Medical Center Corporation | Targeting BCL11A distal regulatory elements for fetal hemoglobin reinduction |
| EP3138911B1 (en) * | 2012-12-06 | 2018-12-05 | Sigma Aldrich Co. LLC | Crispr-based genome modification and regulation |
| ES2576126T3 (es) * | 2012-12-12 | 2016-07-05 | The Broad Institute, Inc. | Modificación por tecnología genética y optimización de sistemas, métodos y composiciones enzimáticas mejorados para la manipulación de secuencias |
| EP3705490B1 (en) | 2012-12-12 | 2024-03-06 | The Broad Institute, Inc. | Engineering and optimization of improved systems, methods and enzyme compositions for sequence manipulation |
| US20140310830A1 (en) | 2012-12-12 | 2014-10-16 | Feng Zhang | CRISPR-Cas Nickase Systems, Methods And Compositions For Sequence Manipulation in Eukaryotes |
| CN119752887A (zh) | 2012-12-12 | 2025-04-04 | 布罗德研究所有限公司 | 用于序列操纵的系统、方法和优化的指导组合物的工程化 |
| WO2014093701A1 (en) * | 2012-12-12 | 2014-06-19 | The Broad Institute, Inc. | Functional genomics using crispr-cas systems, compositions, methods, knock out libraries and applications thereof |
| US8697359B1 (en) * | 2012-12-12 | 2014-04-15 | The Broad Institute, Inc. | CRISPR-Cas systems and methods for altering expression of gene products |
| EP2931892B1 (en) | 2012-12-12 | 2018-09-12 | The Broad Institute, Inc. | Methods, models, systems, and apparatus for identifying target sequences for cas enzymes or crispr-cas systems for target sequences and conveying results thereof |
| DK2931898T3 (en) | 2012-12-12 | 2016-06-20 | Massachusetts Inst Technology | CONSTRUCTION AND OPTIMIZATION OF SYSTEMS, PROCEDURES AND COMPOSITIONS FOR SEQUENCE MANIPULATION WITH FUNCTIONAL DOMAINS |
| US20140179770A1 (en) | 2012-12-12 | 2014-06-26 | Massachusetts Institute Of Technology | Delivery, engineering and optimization of systems, methods and compositions for sequence manipulation and therapeutic applications |
| CN110982844B (zh) * | 2012-12-12 | 2024-08-13 | 布罗德研究所有限公司 | 用于序列操纵的crispr-cas组分系统、方法以及组合物 |
| SG10201704932UA (en) * | 2012-12-17 | 2017-07-28 | Harvard College | Rna-guided human genome engineering |
| WO2014113493A1 (en) | 2013-01-16 | 2014-07-24 | Emory University | Cas9-nucleic acid complexes and uses related thereto |
| WO2014124226A1 (en) | 2013-02-07 | 2014-08-14 | The Rockefeller University | Sequence specific antimicrobials |
| US11135273B2 (en) | 2013-02-07 | 2021-10-05 | The Rockefeller University | Sequence specific antimicrobials |
| CN103981147B (zh) | 2013-02-08 | 2017-11-10 | 中国科学院上海生命科学研究院 | 一种新的制备肝实质细胞的方法 |
| ES2768291T3 (es) * | 2013-02-14 | 2020-06-22 | Univ Osaka | Método para aislar una región genómica específica utilizando una molécula que se une específicamente a una secuencia de ADN endógena |
| CA2900992C (en) | 2013-02-20 | 2023-02-28 | Regeneron Pharmaceuticals, Inc. | Genetic modification of rats |
| EP2922393B2 (en) | 2013-02-27 | 2022-12-28 | Helmholtz Zentrum München - Deutsches Forschungszentrum für Gesundheit und Umwelt (GmbH) | Gene editing in the oocyte by cas9 nucleases |
| ES2749624T3 (es) * | 2013-03-14 | 2020-03-23 | Caribou Biosciences Inc | Composiciones y métodos de ácidos nucleicos dirigidos a ácido nucleico |
| CN108823238B (zh) * | 2013-03-15 | 2022-02-01 | 希博斯美国有限公司 | 采用寡核苷酸介导的基因修复提高靶向基因修饰的效率的方法和组合物 |
| US9234213B2 (en) | 2013-03-15 | 2016-01-12 | System Biosciences, Llc | Compositions and methods directed to CRISPR/Cas genomic engineering systems |
| WO2014204578A1 (en) | 2013-06-21 | 2014-12-24 | The General Hospital Corporation | Using rna-guided foki nucleases (rfns) to increase specificity for rna-guided genome editing |
| US20140273230A1 (en) * | 2013-03-15 | 2014-09-18 | Sigma-Aldrich Co., Llc | Crispr-based genome modification and regulation |
| CA2906747A1 (en) * | 2013-03-15 | 2014-09-18 | Regents Of The University Of Minnesota | Engineering plant genomes using crispr/cas systems |
| US20140349400A1 (en) * | 2013-03-15 | 2014-11-27 | Massachusetts Institute Of Technology | Programmable Modification of DNA |
| US9885033B2 (en) * | 2013-03-15 | 2018-02-06 | The General Hospital Corporation | Increasing specificity for RNA-guided genome editing |
| US10760064B2 (en) * | 2013-03-15 | 2020-09-01 | The General Hospital Corporation | RNA-guided targeting of genetic and epigenomic regulatory proteins to specific genomic loci |
| HK1220864A1 (zh) | 2013-03-15 | 2017-05-19 | 希博斯美国有限公司 | 採用寡核苷酸介導的基因修復提高靶向基因修飾的效率的方法和組合物 |
| US12331303B2 (en) | 2013-03-15 | 2025-06-17 | Cibus Us Llc | Methods and compositions for increasing efficiency of targeted gene modification using oligonucleotide-mediated gene repair |
| US9957515B2 (en) | 2013-03-15 | 2018-05-01 | Cibus Us Llc | Methods and compositions for targeted gene modification |
| US9788534B2 (en) | 2013-03-18 | 2017-10-17 | Kymab Limited | Animal models and therapeutic molecules |
| US9828582B2 (en) | 2013-03-19 | 2017-11-28 | Duke University | Compositions and methods for the induction and tuning of gene expression |
| WO2014172470A2 (en) * | 2013-04-16 | 2014-10-23 | Whitehead Institute For Biomedical Research | Methods of mutating, modifying or modulating nucleic acid in a cell or nonhuman mammal |
| PL3456831T3 (pl) | 2013-04-16 | 2021-12-06 | Regeneron Pharmaceuticals, Inc. | Ukierunkowana modyfikacja genomu szczura |
| US9783618B2 (en) | 2013-05-01 | 2017-10-10 | Kymab Limited | Manipulation of immunoglobulin gene diversity and multi-antibody therapeutics |
| US11707056B2 (en) | 2013-05-02 | 2023-07-25 | Kymab Limited | Animals, repertoires and methods |
| US9783593B2 (en) | 2013-05-02 | 2017-10-10 | Kymab Limited | Antibodies, variable domains and chains tailored for human use |
| JP2016518142A (ja) * | 2013-05-10 | 2016-06-23 | サンガモ バイオサイエンシーズ, インコーポレイテッド | ヌクレアーゼ媒介ゲノム遺伝子操作のための送達方法および組成物 |
| WO2014186435A2 (en) | 2013-05-14 | 2014-11-20 | University Of Georgia Research Foundation, Inc. | Compositions and methods for reducing neointima formation |
| EP3730615A3 (en) | 2013-05-15 | 2020-12-09 | Sangamo Therapeutics, Inc. | Methods and compositions for treatment of a genetic condition |
| WO2014186686A2 (en) * | 2013-05-17 | 2014-11-20 | Two Blades Foundation | Targeted mutagenesis and genome engineering in plants using rna-guided cas nucleases |
| US9873907B2 (en) | 2013-05-29 | 2018-01-23 | Agilent Technologies, Inc. | Method for fragmenting genomic DNA using CAS9 |
| DK3004349T3 (en) * | 2013-05-29 | 2018-06-06 | Cellectis Sa | A method for producing precise DNA cleavage using CAS9 nickase activity |
| US11685935B2 (en) * | 2013-05-29 | 2023-06-27 | Cellectis | Compact scaffold of Cas9 in the type II CRISPR system |
| US9267135B2 (en) * | 2013-06-04 | 2016-02-23 | President And Fellows Of Harvard College | RNA-guided transcriptional regulation |
| EP3603679B1 (en) | 2013-06-04 | 2022-08-10 | President and Fellows of Harvard College | Rna-guided transcriptional regulation |
| KR20230136697A (ko) * | 2013-06-05 | 2023-09-26 | 듀크 유니버시티 | Rna-가이드 유전자 편집 및 유전자 조절 |
| WO2014201015A2 (en) | 2013-06-11 | 2014-12-18 | The Regents Of The University Of California | Methods and compositions for target dna modification |
| CN105531372A (zh) * | 2013-06-14 | 2016-04-27 | 塞尔克蒂斯股份有限公司 | 植物中非转基因基因组编辑方法 |
| KR20160030187A (ko) | 2013-06-17 | 2016-03-16 | 더 브로드 인스티튜트, 인코퍼레이티드 | 간의 표적화 및 치료를 위한 CRISPRCas 시스템, 벡터 및 조성물의 전달 및 용도 |
| KR20160044457A (ko) * | 2013-06-17 | 2016-04-25 | 더 브로드 인스티튜트, 인코퍼레이티드 | 서열 조작을 위한 탠덤 안내 시스템, 방법 및 조성물의 전달, 조작 및 최적화 |
| EP3011030B1 (en) | 2013-06-17 | 2023-11-08 | The Broad Institute, Inc. | Optimized crispr-cas double nickase systems, methods and compositions for sequence manipulation |
| DK3011032T3 (da) | 2013-06-17 | 2020-01-20 | Broad Inst Inc | Fremføring, modificering og optimering af systemer, fremgangsmåder og sammensætninger til målretning mod og modellering af sygdomme og forstyrrelser i postmitotiske celler |
| BR122021009076B1 (pt) | 2013-06-17 | 2024-02-15 | The Broad Institute Inc. | Vetor viral contendo molécula(s) de ácido nucleico heterólogo, composição, uso e métodos do mesmo |
| WO2014204727A1 (en) | 2013-06-17 | 2014-12-24 | The Broad Institute Inc. | Functional genomics using crispr-cas systems, compositions methods, screens and applications thereof |
| JP7120717B2 (ja) * | 2013-07-09 | 2022-08-17 | プレジデント アンド フェローズ オブ ハーバード カレッジ | 多重rna誘導型ゲノム編集 |
| CA3218040A1 (en) | 2013-07-10 | 2015-01-15 | President And Fellows Of Harvard College | Orthogonal cas9 proteins for rna-guided gene regulation and editing |
| US9663782B2 (en) * | 2013-07-19 | 2017-05-30 | Larix Bioscience Llc | Methods and compositions for producing double allele knock outs |
| US10563225B2 (en) | 2013-07-26 | 2020-02-18 | President And Fellows Of Harvard College | Genome engineering |
| US10421957B2 (en) | 2013-07-29 | 2019-09-24 | Agilent Technologies, Inc. | DNA assembly using an RNA-programmable nickase |
| US9944925B2 (en) | 2013-08-02 | 2018-04-17 | Enevolv, Inc. | Processes and host cells for genome, pathway, and biomolecular engineering |
| US20150044192A1 (en) | 2013-08-09 | 2015-02-12 | President And Fellows Of Harvard College | Methods for identifying a target site of a cas9 nuclease |
| WO2015021426A1 (en) * | 2013-08-09 | 2015-02-12 | Sage Labs, Inc. | A crispr/cas system-based novel fusion protein and its application in genome editing |
| US9359599B2 (en) | 2013-08-22 | 2016-06-07 | President And Fellows Of Harvard College | Engineered transcription activator-like effector (TALE) domains and uses thereof |
| WO2015026887A1 (en) | 2013-08-22 | 2015-02-26 | E. I. Du Pont De Nemours And Company | A soybean u6 polymerase iii promoter and methods of use |
| WO2015034872A2 (en) | 2013-09-05 | 2015-03-12 | Massachusetts Institute Of Technology | Tuning microbial populations with programmable nucleases |
| US9388430B2 (en) | 2013-09-06 | 2016-07-12 | President And Fellows Of Harvard College | Cas9-recombinase fusion proteins and uses thereof |
| US9526784B2 (en) | 2013-09-06 | 2016-12-27 | President And Fellows Of Harvard College | Delivery system for functional nucleases |
| US9228207B2 (en) | 2013-09-06 | 2016-01-05 | President And Fellows Of Harvard College | Switchable gRNAs comprising aptamers |
| IL312865B2 (en) | 2013-09-11 | 2025-06-01 | Eagle Biologics Inc | Liquid protein formulations containing viscosity-reducing agents |
| US20150079680A1 (en) | 2013-09-18 | 2015-03-19 | Kymab Limited | Methods, cells & organisms |
| TW201546284A (zh) | 2013-10-01 | 2015-12-16 | Kymab Ltd | 動物模式及治療分子 |
| WO2015065964A1 (en) | 2013-10-28 | 2015-05-07 | The Broad Institute Inc. | Functional genomics using crispr-cas systems, compositions, methods, screens and applications thereof |
| WO2015066119A1 (en) | 2013-10-30 | 2015-05-07 | North Carolina State University | Compositions and methods related to a type-ii crispr-cas system in lactobacillus buchneri |
| WO2015066643A1 (en) * | 2013-11-04 | 2015-05-07 | Dow Agrosciences Llc | Optimal soybean loci |
| US10752906B2 (en) * | 2013-11-05 | 2020-08-25 | President And Fellows Of Harvard College | Precise microbiota engineering at the cellular level |
| WO2015070062A1 (en) * | 2013-11-07 | 2015-05-14 | Massachusetts Institute Of Technology | Cell-based genomic recorded accumulative memory |
| US9834791B2 (en) | 2013-11-07 | 2017-12-05 | Editas Medicine, Inc. | CRISPR-related methods and compositions with governing gRNAS |
| JP2016537028A (ja) * | 2013-11-18 | 2016-12-01 | クリスパー セラピューティクス アーゲー | Crispr−casシステムの材料及び方法 |
| US9074199B1 (en) | 2013-11-19 | 2015-07-07 | President And Fellows Of Harvard College | Mutant Cas9 proteins |
| US10787684B2 (en) * | 2013-11-19 | 2020-09-29 | President And Fellows Of Harvard College | Large gene excision and insertion |
| SMT201800653T1 (it) | 2013-12-11 | 2019-01-11 | Regeneron Pharma | Metodi e composizioni per la modificazione indirizzata a bersaglio di un genoma |
| MX388127B (es) | 2013-12-11 | 2025-03-19 | Regeneron Pharma | Metodos y composiciones para la modificacion dirigida de un genoma. |
| US20150166985A1 (en) * | 2013-12-12 | 2015-06-18 | President And Fellows Of Harvard College | Methods for correcting von willebrand factor point mutations |
| EP3835419A1 (en) * | 2013-12-12 | 2021-06-16 | The Regents of The University of California | Methods and compositions for modifying a single stranded target nucleic acid |
| ES2765481T3 (es) | 2013-12-12 | 2020-06-09 | Broad Inst Inc | Administración, uso y aplicaciones terapéuticas de los sistemas crispr-cas y composiciones para la edición genómica |
| RU2016128077A (ru) | 2013-12-12 | 2018-12-06 | Те Брод Инститьют Инк. | Доставка, применение и применения в терапии систем и композиций crispr-cas для лечения обусловленных hbv и вирусных заболеваний и нарушений |
| WO2015089354A1 (en) * | 2013-12-12 | 2015-06-18 | The Broad Institute Inc. | Compositions and methods of use of crispr-cas systems in nucleotide repeat disorders |
| JP2017503485A (ja) | 2013-12-12 | 2017-02-02 | ザ・ブロード・インスティテュート・インコーポレイテッド | 遺伝子産物の発現、構造情報、及び誘導性モジュラーcas酵素を変更するためのcrispr−cas系並びに方法 |
| EP3079726B1 (en) * | 2013-12-12 | 2018-12-05 | The Broad Institute, Inc. | Delivery, use and therapeutic applications of the crispr-cas systems and compositions for targeting disorders and diseases using particle delivery components |
| WO2015089364A1 (en) | 2013-12-12 | 2015-06-18 | The Broad Institute Inc. | Crystal structure of a crispr-cas system, and uses thereof |
| EP3080271B1 (en) | 2013-12-12 | 2020-02-12 | The Broad Institute, Inc. | Systems, methods and compositions for sequence manipulation with optimized functional crispr-cas systems |
| WO2015086795A1 (en) * | 2013-12-13 | 2015-06-18 | Cellectis | Cas9 nuclease platform for microalgae genome engineering |
| WO2015089375A1 (en) | 2013-12-13 | 2015-06-18 | The General Hospital Corporation | Soluble high molecular weight (hmw) tau species and applications thereof |
| US20150191744A1 (en) * | 2013-12-17 | 2015-07-09 | University Of Massachusetts | Cas9 effector-mediated regulation of transcription, differentiation and gene editing/labeling |
| TWI672379B (zh) | 2013-12-19 | 2019-09-21 | 瑞士商諾華公司 | 人類間皮素嵌合性抗原受體及其用途 |
| EP3083958B1 (en) | 2013-12-19 | 2019-04-17 | Amyris, Inc. | Methods for genomic integration |
| US11672835B2 (en) | 2013-12-20 | 2023-06-13 | Seed Health, Inc. | Method for treating individuals having cancer and who are receiving cancer immunotherapy |
| WO2015095895A1 (en) | 2013-12-20 | 2015-06-25 | Fred Hutchinson Cancer Research Center | Tagged chimeric effector molecules and receptors thereof |
| US11980643B2 (en) | 2013-12-20 | 2024-05-14 | Seed Health, Inc. | Method and system to modify an individual's gut-brain axis to provide neurocognitive protection |
| US11529379B2 (en) | 2013-12-20 | 2022-12-20 | Seed Health, Inc. | Method and system for reducing the likelihood of developing colorectal cancer in an individual human being |
| US11998574B2 (en) | 2013-12-20 | 2024-06-04 | Seed Health, Inc. | Method and system for modulating an individual's skin microbiome |
| US12005085B2 (en) | 2013-12-20 | 2024-06-11 | Seed Health, Inc. | Probiotic method and composition for maintaining a healthy vaginal microbiome |
| US11833177B2 (en) | 2013-12-20 | 2023-12-05 | Seed Health, Inc. | Probiotic to enhance an individual's skin microbiome |
| US12246043B2 (en) | 2013-12-20 | 2025-03-11 | Seed Health, Inc. | Topical application to treat acne vulgaris |
| US11969445B2 (en) | 2013-12-20 | 2024-04-30 | Seed Health, Inc. | Probiotic composition and method for controlling excess weight, obesity, NAFLD and NASH |
| US11642382B2 (en) | 2013-12-20 | 2023-05-09 | Seed Health, Inc. | Method for treating an individual suffering from bladder cancer |
| US11839632B2 (en) | 2013-12-20 | 2023-12-12 | Seed Health, Inc. | Topical application of CRISPR-modified bacteria to treat acne vulgaris |
| US12318377B2 (en) | 2013-12-20 | 2025-06-03 | Seed Health, Inc. | Method and system for reducing the likelihood of a porphyromonas gingivalis infection in a human being |
| US12329783B2 (en) | 2013-12-20 | 2025-06-17 | Seed Health, Inc. | Method and system to improve the health of a person's skin microbiome |
| US11826388B2 (en) | 2013-12-20 | 2023-11-28 | Seed Health, Inc. | Topical application of Lactobacillus crispatus to ameliorate barrier damage and inflammation |
| EP3087101B1 (en) | 2013-12-20 | 2024-06-05 | Novartis AG | Regulatable chimeric antigen receptor |
| US11213552B2 (en) | 2015-11-30 | 2022-01-04 | Joseph E. Kovarik | Method for treating an individual suffering from a chronic infectious disease and cancer |
| US11026982B2 (en) | 2015-11-30 | 2021-06-08 | Joseph E. Kovarik | Method for reducing the likelihood of developing bladder or colorectal cancer in an individual human being |
| AU2014370416B2 (en) * | 2013-12-26 | 2021-03-11 | The General Hospital Corporation | Multiplex guide RNAs |
| EP3089989B1 (en) * | 2013-12-31 | 2020-06-24 | The Regents of The University of California | Cas9 crystals and methods of use thereof |
| CN106133141B (zh) * | 2014-01-08 | 2021-08-20 | 哈佛学院董事及会员团体 | Rna引导的基因驱动 |
| JP2017502683A (ja) * | 2014-01-14 | 2017-01-26 | ラム セラピューティクス, インコーポレイテッド | 変異誘発方法 |
| US10787654B2 (en) | 2014-01-24 | 2020-09-29 | North Carolina State University | Methods and compositions for sequence guiding Cas9 targeting |
| WO2015112896A2 (en) * | 2014-01-24 | 2015-07-30 | North Carolina State University | Methods and compositions for sequences guiding cas9 targeting |
| US11315659B2 (en) * | 2014-01-27 | 2022-04-26 | Georgia Tech Research Corporation | Methods and systems for identifying nucleotide-guided nuclease off-target sites |
| US10354746B2 (en) * | 2014-01-27 | 2019-07-16 | Georgia Tech Research Corporation | Methods and systems for identifying CRISPR/Cas off-target sites |
| WO2015116686A1 (en) * | 2014-01-29 | 2015-08-06 | Agilent Technologies, Inc. | Cas9-based isothermal method of detection of specific dna sequence |
| EP3795697B1 (en) * | 2014-02-04 | 2024-09-18 | Jumpcode Genomics, Inc. | Genome fractioning |
| HRP20201040T1 (hr) | 2014-02-11 | 2021-01-08 | The Regents Of The University Of Colorado, A Body Corporate | Inženjering složenog genoma omogućen sustavom crispr |
| US20180142307A1 (en) * | 2014-02-11 | 2018-05-24 | California Institute Of Technology | Recording and mapping lineage information and molecular events in individual cells |
| US10287590B2 (en) * | 2014-02-12 | 2019-05-14 | Dna2.0, Inc. | Methods for generating libraries with co-varying regions of polynuleotides for genome modification |
| EP3105325B2 (en) | 2014-02-13 | 2024-10-23 | Takara Bio USA, Inc. | Methods of depleting a target molecule from an initial collection of nucleic acids, and compositions and kits for practicing the same |
| SG11201606819QA (en) | 2014-02-18 | 2016-09-29 | Univ Duke | Compositions for the inactivation of virus replication and methods of making and using the same |
| WO2015127439A1 (en) * | 2014-02-24 | 2015-08-27 | Sangamo Biosciences, Inc. | Methods and compositions for nuclease-mediated targeted integration |
| CA2940217C (en) | 2014-02-27 | 2023-06-13 | Monsanto Technology Llc | Compositions and methods for site directed genomic modification |
| CN103820454B (zh) * | 2014-03-04 | 2016-03-30 | 上海金卫生物技术有限公司 | CRISPR-Cas9特异性敲除人PD1基因的方法以及用于特异性靶向PD1基因的sgRNA |
| CN111500570B (zh) * | 2014-03-05 | 2025-06-20 | 国立大学法人神户大学 | 特异性转变靶向dna序列的核酸碱基的基因组序列的修饰方法、及其使用的分子复合体 |
| EP3957735A1 (en) | 2014-03-05 | 2022-02-23 | Editas Medicine, Inc. | Crispr/cas-related methods and compositions for treating usher syndrome and retinitis pigmentosa |
| US11141493B2 (en) | 2014-03-10 | 2021-10-12 | Editas Medicine, Inc. | Compositions and methods for treating CEP290-associated disease |
| US9938521B2 (en) | 2014-03-10 | 2018-04-10 | Editas Medicine, Inc. | CRISPR/CAS-related methods and compositions for treating leber's congenital amaurosis 10 (LCA10) |
| US11339437B2 (en) | 2014-03-10 | 2022-05-24 | Editas Medicine, Inc. | Compositions and methods for treating CEP290-associated disease |
| PT3116305T (pt) | 2014-03-14 | 2024-03-07 | Cibus Us Llc | Métodos e composições para aumentar a eficiência da modificação genética direccionada utilizando a reparação genética mediada por oligonucleótidos |
| EP3119425B1 (en) | 2014-03-15 | 2020-09-23 | Novartis AG | Regulatable chimeric antigen receptor |
| ES2939760T3 (es) | 2014-03-15 | 2023-04-26 | Novartis Ag | Tratamiento de cáncer utilizando un receptor quimérico para antígenos |
| EP3628334B1 (en) | 2014-03-21 | 2023-06-28 | Genzyme Corporation | Gene therapy for retinitis pigmentosa |
| US10451633B2 (en) * | 2014-03-24 | 2019-10-22 | IMMCO Diagnostics, Inc. | Anti-nuclear antibody detection and diagnostics for systemic and non-systemic autoimmune disorders |
| JP2017512500A (ja) * | 2014-03-25 | 2017-05-25 | ギンゴー バイオワークス, インコーポレイテッド | 細胞工学のための方法および遺伝システム |
| EP3122880B1 (en) | 2014-03-26 | 2021-05-05 | Editas Medicine, Inc. | Crispr/cas-related methods and compositions for treating sickle cell disease |
| US11318206B2 (en) | 2014-03-28 | 2022-05-03 | Aposense Ltd | Compounds and methods for trans-membrane delivery of molecules |
| CA2944141C (en) * | 2014-03-28 | 2023-03-28 | Aposense Ltd. | Compounds and methods for trans-membrane delivery of molecules |
| WO2015153791A1 (en) * | 2014-04-01 | 2015-10-08 | Editas Medicine, Inc. | Crispr/cas-related methods and compositions for treating herpes simplex virus type 2 (hsv-2) |
| EP3126503A1 (en) * | 2014-04-03 | 2017-02-08 | Massachusetts Institute Of Technology | Methods and compositions for the production of guide rna |
| DK3888674T3 (da) | 2014-04-07 | 2024-07-08 | Novartis Ag | Behandling af cancer ved anvendelse af anti-cd19-kimær antigenreceptor |
| CA2944978C (en) | 2014-04-08 | 2024-02-13 | North Carolina State University | Methods and compositions for rna-directed repression of transcription using crispr-associated genes |
| CA2945335A1 (en) * | 2014-04-18 | 2015-10-22 | Editas Medicine, Inc. | Crispr-cas-related methods, compositions and components for cancer immunotherapy |
| SG10201809290SA (en) | 2014-04-25 | 2019-01-30 | Childrens Medical Ct Corp | Compositions and Methods to Treating Hemoglobinopathies |
| CN106535630B (zh) | 2014-04-28 | 2020-04-24 | 重组股份有限公司 | 多重基因编辑 |
| WO2015168404A1 (en) * | 2014-04-30 | 2015-11-05 | Massachusetts Institute Of Technology | Toehold-gated guide rna for programmable cas9 circuitry with rna input |
| PL3140269T3 (pl) | 2014-05-09 | 2024-03-11 | Yale University | Cząstki pokryte hiperrozgałęzionym poliglicerolem oraz sposoby ich wytwarzania i zastosowania |
| US11918695B2 (en) | 2014-05-09 | 2024-03-05 | Yale University | Topical formulation of hyperbranched polymer-coated particles |
| WO2015175642A2 (en) * | 2014-05-13 | 2015-11-19 | Sangamo Biosciences, Inc. | Methods and compositions for prevention or treatment of a disease |
| DK3145934T5 (da) | 2014-05-19 | 2020-11-30 | Pfizer | Substituerede-6,8-dioxabicyclo[3.2.1]octan-2,3-diol-forbindelser som ASGPR-targeteringsmidler |
| JP2017517256A (ja) * | 2014-05-20 | 2017-06-29 | リージェンツ オブ ザ ユニバーシティ オブ ミネソタ | 遺伝子配列を編集する方法 |
| CN106661548B (zh) | 2014-05-28 | 2020-12-11 | 儿童医院医疗中心 | 用于经由定向分化将前体细胞转化为胃组织的方法和系统 |
| JP2017518075A (ja) | 2014-05-30 | 2017-07-06 | ザ ボード オブ トラスティーズ オブ ザ レランド スタンフォード ジュニア ユニバーシティー | 潜伏性ウイルス感染を処置するための組成物および方法 |
| EP3152319A4 (en) * | 2014-06-05 | 2017-12-27 | Sangamo BioSciences, Inc. | Methods and compositions for nuclease design |
| WO2015188109A1 (en) | 2014-06-06 | 2015-12-10 | Regeneron Pharmaceuticals, Inc. | Methods and compositions for modifying a targeted locus |
| EP3155116A4 (en) | 2014-06-10 | 2017-12-27 | Massachusetts Institute Of Technology | Method for gene editing |
| US11274302B2 (en) * | 2016-08-17 | 2022-03-15 | Diacarta Ltd | Specific synthetic chimeric Xenonucleic acid guide RNA; s(XNA-gRNA) for enhancing CRISPR mediated genome editing efficiency |
| AU2015324564B2 (en) | 2014-06-11 | 2021-07-01 | Duke University | Compositions and methods for rapid and dynamic flux control using synthetic metabolic valves |
| WO2015192020A1 (en) | 2014-06-13 | 2015-12-17 | Children's Medical Center Corporation | Products and methods to isolate mitochondria |
| US20170107541A1 (en) * | 2014-06-17 | 2017-04-20 | Poseida Therapeutics, Inc. | A method for directing proteins to specific loci in the genome and uses thereof |
| NZ765591A (en) | 2014-06-23 | 2022-09-30 | Regeneron Pharma | Nuclease-mediated dna assembly |
| EP3919621A1 (en) | 2014-06-23 | 2021-12-08 | The General Hospital Corporation | Genomewide unbiased identification of dsbs evaluated by sequencing (guide-seq) |
| US9902971B2 (en) | 2014-06-26 | 2018-02-27 | Regeneron Pharmaceuticals, Inc. | Methods for producing a mouse XY embryonic (ES) cell line capable of producing a fertile XY female mouse in an F0 generation |
| CN106687594A (zh) | 2014-07-11 | 2017-05-17 | 纳幕尔杜邦公司 | 用于产生对草甘膦除草剂具有抗性的植物的组合物和方法 |
| US10179932B2 (en) | 2014-07-11 | 2019-01-15 | President And Fellows Of Harvard College | Methods for high-throughput labelling and detection of biological features in situ using microscopy |
| US20170219596A1 (en) * | 2014-07-14 | 2017-08-03 | The Regents Of The University Of California | A protein tagging system for in vivo single molecule imaging and control of gene transcription |
| WO2016011080A2 (en) * | 2014-07-14 | 2016-01-21 | The Regents Of The University Of California | Crispr/cas transcriptional modulation |
| AU2015289644A1 (en) | 2014-07-15 | 2017-02-02 | Juno Therapeutics, Inc. | Engineered cells for adoptive cell therapy |
| US20160053304A1 (en) * | 2014-07-18 | 2016-02-25 | Whitehead Institute For Biomedical Research | Methods Of Depleting Target Sequences Using CRISPR |
| US20160053272A1 (en) * | 2014-07-18 | 2016-02-25 | Whitehead Institute For Biomedical Research | Methods Of Modifying A Sequence Using CRISPR |
| EP3172237A2 (en) | 2014-07-21 | 2017-05-31 | Novartis AG | Treatment of cancer using humanized anti-bcma chimeric antigen receptor |
| WO2016014553A1 (en) | 2014-07-21 | 2016-01-28 | Novartis Ag | Sortase synthesized chimeric antigen receptors |
| SG11201702066UA (en) * | 2014-07-21 | 2017-04-27 | Illumina Inc | Polynucleotide enrichment using crispr-cas systems |
| CA2955154C (en) | 2014-07-21 | 2023-10-31 | Novartis Ag | Treatment of cancer using a cd33 chimeric antigen receptor |
| EP3193915A1 (en) | 2014-07-21 | 2017-07-26 | Novartis AG | Combinations of low, immune enhancing. doses of mtor inhibitors and cars |
| EP4079847A1 (en) * | 2014-07-30 | 2022-10-26 | President And Fellows Of Harvard College | Cas9 proteins including ligand-dependent inteins |
| EP3660042B1 (en) | 2014-07-31 | 2023-01-11 | Novartis AG | Subset-optimized chimeric antigen receptor-containing t-cells |
| US20160076093A1 (en) * | 2014-08-04 | 2016-03-17 | University Of Washington | Multiplex homology-directed repair |
| CN113789317B (zh) | 2014-08-06 | 2024-02-23 | 基因工具股份有限公司 | 使用空肠弯曲杆菌crispr/cas系统衍生的rna引导的工程化核酸酶的基因编辑 |
| US9932566B2 (en) * | 2014-08-07 | 2018-04-03 | Agilent Technologies, Inc. | CIS-blocked guide RNA |
| US10513711B2 (en) | 2014-08-13 | 2019-12-24 | Dupont Us Holding, Llc | Genetic targeting in non-conventional yeast using an RNA-guided endonuclease |
| WO2016025759A1 (en) | 2014-08-14 | 2016-02-18 | Shen Yuelei | Dna knock-in system |
| CA2958200A1 (en) | 2014-08-14 | 2016-02-18 | Novartis Ag | Treatment of cancer using a gfr alpha-4 chimeric antigen receptor |
| US9879270B2 (en) * | 2014-08-15 | 2018-01-30 | Wisconsin Alumni Research Foundation | Constructs and methods for genome editing and genetic engineering of fungi and protists |
| EP3712171A1 (en) | 2014-08-19 | 2020-09-23 | Novartis AG | Treatment of cancer using a cd123 chimeric antigen receptor |
| EP3183367B1 (en) | 2014-08-19 | 2019-06-26 | Pacific Biosciences Of California, Inc. | Compositions and methods for enrichment of nucleic acids |
| US10435685B2 (en) | 2014-08-19 | 2019-10-08 | Pacific Biosciences Of California, Inc. | Compositions and methods for enrichment of nucleic acids |
| KR20170036801A (ko) | 2014-08-19 | 2017-04-03 | 프레지던트 앤드 펠로우즈 오브 하바드 칼리지 | 핵산의 프로빙 및 맵핑을 위한 rna-가이드된 시스템 |
| DK3186376T3 (da) | 2014-08-27 | 2019-05-06 | Caribou Biosciences Inc | Fremgangsmåder til forbedring af cas9-medieret manipulationseffektivitet |
| EP3186375A4 (en) * | 2014-08-28 | 2019-03-13 | North Carolina State University | Novel CAS9 PROTEINS AND CHARACTERISTICS FOR DNA TARGETING AND GENOME EDITING |
| EP3188763B1 (en) | 2014-09-02 | 2020-05-13 | The Regents of The University of California | Methods and compositions for rna-directed target dna modification |
| RU2017112324A (ru) | 2014-09-12 | 2018-10-15 | Пайонир Хай-Бред Интернэшнл, Инк. | Создание сайтов сайт-специфической интеграции для сложных локусов признаков в кукурузе и сое, а также способы применения |
| KR102804360B1 (ko) | 2014-09-17 | 2025-05-12 | 노파르티스 아게 | 입양 면역요법을 위한 키메라 수용체에 의한 세포독성 세포의 표적화 |
| WO2016049024A2 (en) * | 2014-09-24 | 2016-03-31 | The Broad Institute Inc. | Delivery, use and therapeutic applications of the crispr-cas systems and compositions for modeling competition of multiple cancer mutations in vivo |
| CN107205353B (zh) | 2014-09-26 | 2021-03-19 | 谱赛科美国股份有限公司 | 用于甜菊的单核苷酸多态性(snp)标志物 |
| US20170233762A1 (en) * | 2014-09-29 | 2017-08-17 | The Regents Of The University Of California | Scaffold rnas |
| CN107002098A (zh) | 2014-09-29 | 2017-08-01 | 杰克逊实验室 | 通过电穿孔高效率、高通量产生遗传修饰哺乳动物 |
| JP6716577B2 (ja) | 2014-10-01 | 2020-07-01 | イーグル・バイオロジクス・インコーポレイテッドEagle Biologics, Inc. | 粘度低下物質を含有する、多糖および核酸の調合薬 |
| WO2016050512A1 (en) | 2014-10-03 | 2016-04-07 | Bayer Cropscience Nv | Methods and means for increasing stress tolerance and biomass in plants |
| EP3204496A1 (en) | 2014-10-10 | 2017-08-16 | Editas Medicine, Inc. | Compositions and methods for promoting homology directed repair |
| EP3561052A1 (en) | 2014-10-15 | 2019-10-30 | Regeneron Pharmaceuticals, Inc. | Methods and compositions for generating or maintaining pluripotent cells |
| EP3207131B1 (en) * | 2014-10-17 | 2022-09-28 | Howard Hughes Medical Institute | Genomic probes |
| US20170306306A1 (en) * | 2014-10-24 | 2017-10-26 | Life Technologies Corporation | Compositions and Methods for Enhancing Homologous Recombination |
| GB201418965D0 (ko) | 2014-10-24 | 2014-12-10 | Ospedale San Raffaele And Fond Telethon | |
| KR102546296B1 (ko) | 2014-10-31 | 2023-06-21 | 더 트러스티스 오브 더 유니버시티 오브 펜실베니아 | 변형된 t 세포에서 유전자 발현의 변경 및 그의 용도 |
| CN107429246B (zh) * | 2014-10-31 | 2021-06-01 | 麻省理工学院 | 用于crispr的大规模并行组合遗传学 |
| US20180291382A1 (en) * | 2014-11-05 | 2018-10-11 | The Regents Of The University Of California | Methods for Autocatalytic Genome Editing and Neutralizing Autocatalytic Genome Editing |
| CN107406838A (zh) * | 2014-11-06 | 2017-11-28 | 纳幕尔杜邦公司 | Rna引导的内切核酸酶向细胞中的肽介导的递送 |
| US11680268B2 (en) | 2014-11-07 | 2023-06-20 | Editas Medicine, Inc. | Methods for improving CRISPR/Cas-mediated genome-editing |
| WO2016080097A1 (ja) * | 2014-11-17 | 2016-05-26 | 国立大学法人東京医科歯科大学 | 簡便で高効率の遺伝子改変非ヒト哺乳動物の作製方法 |
| CN104531632A (zh) * | 2014-11-18 | 2015-04-22 | 李云英 | 快速降解的Cas9-ODC422-461融合蛋白及其应用 |
| EP3521437B1 (en) | 2014-11-21 | 2025-01-15 | Regeneron Pharmaceuticals, Inc. | Methods for targeted genetic modification using paired guide rnas |
| CN107208070B (zh) * | 2014-11-26 | 2021-09-07 | 技术创新动力基金(以色列)有限合伙公司 | 细菌基因的靶向消除 |
| GB201421096D0 (en) | 2014-11-27 | 2015-01-14 | Imp Innovations Ltd | Genome editing methods |
| US10479997B2 (en) | 2014-12-01 | 2019-11-19 | Novartis Ag | Compositions and methods for diagnosis and treatment of prostate cancer |
| AU2015355546B2 (en) | 2014-12-03 | 2021-10-14 | Agilent Technologies, Inc. | Guide RNA with chemical modifications |
| US9888673B2 (en) | 2014-12-10 | 2018-02-13 | Regents Of The University Of Minnesota | Genetically modified cells, tissues, and organs for treating disease |
| EP3985115A1 (en) | 2014-12-12 | 2022-04-20 | The Broad Institute, Inc. | Protected guide rnas (pgrnas) |
| EP3230445B1 (en) * | 2014-12-12 | 2024-01-24 | Tod M. Woolf | Compositions and methods for editing nucleic acids in cells utilizing oligonucleotides |
| WO2016094874A1 (en) * | 2014-12-12 | 2016-06-16 | The Broad Institute Inc. | Escorted and functionalized guides for crispr-cas systems |
| WO2016093668A2 (ko) * | 2014-12-12 | 2016-06-16 | 한국한의학연구원 | 일체형 유전자 치료 유도만능줄기세포 제작방법 |
| CN107278227B (zh) | 2014-12-16 | 2021-05-28 | C3J治疗公司 | 用于体外病毒基因组工程的组合物和方法 |
| US20170369866A1 (en) * | 2014-12-17 | 2017-12-28 | E I Du Pont De Nemours And Company | Compositions and methods for efficient gene editing in e. coli using guide rna/cas endonuclease systems in combination with circular polynucleotide modification templates |
| WO2016097751A1 (en) * | 2014-12-18 | 2016-06-23 | The University Of Bath | Method of cas9 mediated genome engineering |
| EP3786296A1 (en) | 2014-12-18 | 2021-03-03 | Integrated Dna Technologies, Inc. | Crispr-based compositions and methods of use |
| WO2016100857A1 (en) | 2014-12-19 | 2016-06-23 | Regeneron Pharmaceuticals, Inc. | Stem cells for modeling type 2 diabetes |
| MX388784B (es) | 2014-12-19 | 2025-03-20 | Regeneron Pharma | Métodos y composiciones para la modificación genética dirigida a través de direccionamiento múltiple de una sola etapa. |
| WO2016098078A2 (en) | 2014-12-19 | 2016-06-23 | Novartis Ag | Dimerization switches and uses thereof |
| EP3957745A1 (en) | 2014-12-20 | 2022-02-23 | Arc Bio, LLC | Compositions and methods for targeted depletion, enrichment, and partitioning of nucleic acids using crispr/cas system proteins |
| CA2969151A1 (en) | 2014-12-23 | 2016-06-30 | Syngenta Participations Ag | Methods and compositions for identifying and enriching for cells comprising site specific genomic modifications |
| CA2970370A1 (en) * | 2014-12-24 | 2016-06-30 | Massachusetts Institute Of Technology | Crispr having or associated with destabilization domains |
| EP3240889A4 (en) * | 2014-12-31 | 2018-06-20 | Synthetic Genomics, Inc. | Compositions and methods for high efficiency in vivo genome editing |
| WO2016110453A1 (en) * | 2015-01-06 | 2016-07-14 | Dsm Ip Assets B.V. | A crispr-cas system for a filamentous fungal host cell |
| WO2016114972A1 (en) | 2015-01-12 | 2016-07-21 | The Regents Of The University Of California | Heterodimeric cas9 and methods of use thereof |
| MA41349A (fr) * | 2015-01-14 | 2017-11-21 | Univ Temple | Éradication de l'herpès simplex de type i et d'autres virus de l'herpès associés guidée par arn |
| US10059940B2 (en) * | 2015-01-27 | 2018-08-28 | Minghong Zhong | Chemically ligated RNAs for CRISPR/Cas9-lgRNA complexes as antiviral therapeutic agents |
| CN105821073B (zh) | 2015-01-27 | 2021-08-10 | 中国科学院遗传与发育生物学研究所 | 一种通过基因瞬时表达对整株植物定点改造的方法 |
| WO2016123243A1 (en) | 2015-01-28 | 2016-08-04 | The Regents Of The University Of California | Methods and compositions for labeling a single-stranded target nucleic acid |
| SG11201705788TA (en) | 2015-01-28 | 2017-08-30 | Pioneer Hi Bred Int | Crispr hybrid dna/rna polynucleotides and methods of use |
| HK1248755A1 (zh) * | 2015-01-30 | 2018-10-19 | The Regents Of The University Of California | 主要造血細胞中的蛋白質傳遞 |
| KR102699584B1 (ko) * | 2015-02-02 | 2024-08-28 | 메이라지티엑스 유케이 Ii 리미티드 | 대안적 스플라이싱의 압타머 매개 조절에 의한 유전자 발현 제어 |
| AU2016216149B2 (en) | 2015-02-06 | 2022-03-31 | National University Of Singapore | Methods for enhancing efficacy of therapeutic immune cells |
| US10676726B2 (en) | 2015-02-09 | 2020-06-09 | Duke University | Compositions and methods for epigenome editing |
| WO2016133165A1 (ja) * | 2015-02-19 | 2016-08-25 | 国立大学法人徳島大学 | Cas9 mRNAを哺乳動物の受精卵にエレクトロポレーションにより導入する方法 |
| AU2016225179C1 (en) | 2015-02-23 | 2022-11-03 | Crispr Therapeutics Ag | Materials and methods for treatment of hemoglobinopathies |
| WO2016135559A2 (en) | 2015-02-23 | 2016-09-01 | Crispr Therapeutics Ag | Materials and methods for treatment of human genetic diseases including hemoglobinopathies |
| WO2016137949A1 (en) * | 2015-02-23 | 2016-09-01 | Voyager Therapeutics, Inc. | Regulatable expression using adeno-associated virus (aav) |
| US10968536B2 (en) | 2015-02-25 | 2021-04-06 | Jumpcode Genomics, Inc. | Methods and compositions for sequencing |
| WO2016142427A1 (en) | 2015-03-10 | 2016-09-15 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Method ank kit for reprogramming somatic cells |
| KR102004076B1 (ko) | 2015-03-13 | 2019-07-25 | 더 잭슨 래보라토리 | 3-성분 crispr/cas 복합체 시스템 및 그 용도 |
| EP3279321A4 (en) | 2015-03-16 | 2018-10-31 | Institute Of Genetics And Developmental Biology, Chinese Academy Of Sciences | Method of applying non-genetic substance to perform site-directed reform of plant genome |
| US11046952B2 (en) * | 2015-03-16 | 2021-06-29 | The Broad Institute, Inc. | Encoding of DNA vector identity via iterative hybridization detection of a barcode transcript |
| US10066256B2 (en) | 2015-03-17 | 2018-09-04 | Bio-Rad Laboratories, Inc. | Detection of genome editing |
| ES2860712T3 (es) | 2015-03-24 | 2021-10-05 | Univ California | Variantes de virus adenoasociados y métodos de uso de las mismas |
| US20180112213A1 (en) * | 2015-03-25 | 2018-04-26 | Editas Medicine, Inc. | Crispr/cas-related methods, compositions and components |
| EP3274460A1 (en) | 2015-03-27 | 2018-01-31 | E. I. du Pont de Nemours and Company | Soybean u6 small nuclear rna gene promoters and their use in constitutive expression of small rna genes in plants |
| CN108124453B (zh) | 2015-03-31 | 2022-04-05 | 爱克莱根科技公司 | 用于将DNA序列靶向并入细胞或生物体的基因组中的Cas9逆转录病毒整合酶和Cas9重组酶系统 |
| EP3280803B1 (en) | 2015-04-06 | 2021-05-26 | The Board of Trustees of the Leland Stanford Junior University | Chemically modified guide rnas for crispr/cas-mediated gene regulation |
| WO2016167300A1 (ja) * | 2015-04-13 | 2016-10-20 | 国立大学法人東京大学 | 光依存的に又は薬物存在下でヌクレアーゼ活性若しくはニッカーゼ活性を示す、又は標的遺伝子の発現を抑制若しくは活性化するポリペプチドのセット |
| US10544424B2 (en) | 2015-04-15 | 2020-01-28 | Synthetic Genomics, Inc. | Algal chloroplastic SRP54 mutants |
| US11674144B2 (en) * | 2015-04-16 | 2023-06-13 | California Institute Of Technology | Fractional regulation of transcription |
| WO2016172583A1 (en) | 2015-04-23 | 2016-10-27 | Novartis Ag | Treatment of cancer using chimeric antigen receptor and protein kinase a blocker |
| EP3286571B1 (en) * | 2015-04-24 | 2021-08-18 | Editas Medicine, Inc. | Evaluation of cas9 molecule/guide rna molecule complexes |
| WO2016176652A2 (en) | 2015-04-29 | 2016-11-03 | Fred Hutchinson Cancer Research Center | Modified stem cells and uses thereof |
| WO2016178207A1 (en) * | 2015-05-04 | 2016-11-10 | Ramot At Tel-Aviv University Ltd. | Methods and kits for fragmenting dna |
| EP3461337A1 (en) | 2015-05-06 | 2019-04-03 | Snipr Technologies Limited | Altering microbial populations & modifying microbiota |
| JP7288302B2 (ja) * | 2015-05-08 | 2023-06-07 | ザ チルドレンズ メディカル センター コーポレーション | 胎児型ヘモグロビン再誘導のための、bcl11aエンハンサー機能性領域を標的とする方法 |
| US11253616B2 (en) | 2017-09-06 | 2022-02-22 | The Trustees Of The University Of Pennsylvania | Small molecules for dual function positron emission tomography (PET) and cell suicide switches |
| US11390884B2 (en) | 2015-05-11 | 2022-07-19 | Editas Medicine, Inc. | Optimized CRISPR/cas9 systems and methods for gene editing in stem cells |
| WO2016183402A2 (en) * | 2015-05-13 | 2016-11-17 | President And Fellows Of Harvard College | Methods of making and using guide rna for use with cas9 systems |
| EP3294879A4 (en) * | 2015-05-14 | 2019-02-20 | University of Southern California | OPTIMIZED GENETIZATION WITH A RECOMBINANT ENDONUCLEASE SYSTEM |
| WO2016186946A1 (en) * | 2015-05-15 | 2016-11-24 | Pioneer Hi-Bred International, Inc. | Rapid characterization of cas endonuclease systems, pam sequences and guide rna elements |
| WO2016186745A1 (en) * | 2015-05-15 | 2016-11-24 | Ge Healthcare Dharmacon, Inc. | Synthetic single guide rna for cas9-mediated gene editing |
| EP3095870A1 (en) | 2015-05-19 | 2016-11-23 | Kws Saat Se | Methods for the in planta transformation of plants and manufacturing processes and products based and obtainable therefrom |
| US10966414B2 (en) | 2015-05-26 | 2021-04-06 | California Institute Of Technology | Population control using engineered translocations |
| HK1253712A1 (zh) | 2015-05-29 | 2019-06-28 | North Carolina State University | 使用crispr核酸篩選細菌、古細菌、藻類和酵母的方法 |
| US10117911B2 (en) | 2015-05-29 | 2018-11-06 | Agenovir Corporation | Compositions and methods to treat herpes simplex virus infections |
| UY36703A (es) | 2015-06-02 | 2016-12-30 | Monsanto Technology Llc | Composiciones y métodos para la administración de un polinucleótido en una planta |
| US10392607B2 (en) | 2015-06-03 | 2019-08-27 | The Regents Of The University Of California | Cas9 variants and methods of use thereof |
| CN108368502B (zh) * | 2015-06-03 | 2022-03-18 | 内布拉斯加大学董事委员会 | 使用单链dna的dna编辑 |
| US20180296537A1 (en) | 2015-06-05 | 2018-10-18 | Novartis Ag | Methods and compositions for diagnosing, treating, and monitoring treatment of shank3 deficiency associated disorders |
| KR102796744B1 (ko) | 2015-06-09 | 2025-04-15 | 에디타스 메디신, 인코포레이티드 | 이식의 개선을 위한 crispr/cas-관련 방법 및 조성물 |
| US20160362667A1 (en) | 2015-06-10 | 2016-12-15 | Caribou Biosciences, Inc. | CRISPR-Cas Compositions and Methods |
| GB201510296D0 (en) * | 2015-06-12 | 2015-07-29 | Univ Wageningen | Thermostable CAS9 nucleases |
| KR20180034389A (ko) | 2015-06-12 | 2018-04-04 | 론자 워커스빌 아이엔씨. | 합성 전사인자를 이용한 핵 역분화 방법 |
| CA2983874C (en) | 2015-06-15 | 2022-06-21 | North Carolina State University | Methods and compositions for efficient delivery of nucleic acids and rna-based antimicrobials |
| IL316159A (en) | 2015-06-15 | 2024-12-01 | Mpeg La Llc | Defined Oligonucleotide Multimers and Methods for Manufacture Thereof |
| US11555207B2 (en) | 2015-06-17 | 2023-01-17 | The Uab Research Foundation | CRISPR/Cas9 complex for introducing a functional polypeptide into cells of blood cell lineage |
| CA2989831A1 (en) | 2015-06-17 | 2016-12-22 | The Uab Research Foundation | Crispr/cas9 complex for genomic editing |
| PT3310909T (pt) * | 2015-06-17 | 2021-09-09 | Poseida Therapeutics Inc | Composições e métodos para dirigir proteínas para loci específicos no genoma |
| AU2016279062A1 (en) * | 2015-06-18 | 2019-03-28 | Omar O. Abudayyeh | Novel CRISPR enzymes and systems |
| US9790490B2 (en) | 2015-06-18 | 2017-10-17 | The Broad Institute Inc. | CRISPR enzymes and systems |
| WO2016205759A1 (en) | 2015-06-18 | 2016-12-22 | The Broad Institute Inc. | Engineering and optimization of systems, methods, enzymes and guide scaffolds of cas9 orthologs and variants for sequence manipulation |
| WO2016205613A1 (en) | 2015-06-18 | 2016-12-22 | The Broad Institute Inc. | Crispr enzyme mutations reducing off-target effects |
| AU2016285724A1 (en) | 2015-06-29 | 2017-11-16 | Ionis Pharmaceuticals, Inc. | Modified CRISPR RNA and modified single CRISPR RNA and uses thereof |
| WO2017004279A2 (en) * | 2015-06-29 | 2017-01-05 | Massachusetts Institute Of Technology | Compositions comprising nucleic acids and methods of using the same |
| JP6765665B2 (ja) * | 2015-07-13 | 2020-10-07 | 国立研究開発法人農業・食品産業技術総合研究機構 | 不稔化植物、不稔化植物の作出方法、及びベクター |
| CN108291218B (zh) | 2015-07-15 | 2022-08-19 | 新泽西鲁特格斯州立大学 | 核酸酶非依赖性靶向基因编辑平台及其用途 |
| MA42895A (fr) | 2015-07-15 | 2018-05-23 | Juno Therapeutics Inc | Cellules modifiées pour thérapie cellulaire adoptive |
| WO2017015101A1 (en) * | 2015-07-17 | 2017-01-26 | University Of Washington | Methods for maximizing the efficiency of targeted gene correction |
| US10676735B2 (en) | 2015-07-22 | 2020-06-09 | Duke University | High-throughput screening of regulatory element function with epigenome editing technologies |
| WO2017023801A1 (en) | 2015-07-31 | 2017-02-09 | Regents Of The University Of Minnesota | Intracellular genomic transplant and methods of therapy |
| WO2017024047A1 (en) * | 2015-08-03 | 2017-02-09 | Emendobio Inc. | Compositions and methods for increasing nuclease induced recombination rate in cells |
| EP3331906A1 (en) | 2015-08-06 | 2018-06-13 | Dana-Farber Cancer Institute, Inc. | Tunable endogenous protein degradation |
| US9580727B1 (en) | 2015-08-07 | 2017-02-28 | Caribou Biosciences, Inc. | Compositions and methods of engineered CRISPR-Cas9 systems using split-nexus Cas9-associated polynucleotides |
| AU2016306275A1 (en) | 2015-08-07 | 2018-02-08 | Arrowhead Pharmaceuticals, Inc. | RNAi therapy for Hepatitis B virus infection |
| EP3331913A1 (en) | 2015-08-07 | 2018-06-13 | Novartis AG | Treatment of cancer using chimeric cd3 receptor proteins |
| EP3334746B1 (en) | 2015-08-14 | 2021-11-24 | The University Of Sydney | Connexin 45 inhibition for therapy |
| KR20200078685A (ko) | 2015-08-14 | 2020-07-01 | 인스티튜트 오브 제네틱스 앤드 디벨롭멘털 바이오롤지, 차이니즈 아카데미 오브 사이언시스 | 부위-특이적인 뉴클레오티드 치환을 통해 글리포세이트-내성 벼를 수득하는 방법 |
| US10898522B2 (en) | 2015-08-19 | 2021-01-26 | Children's Research Institute, Children's National Medical Center | Compositions and methods for treating graft versus host disease |
| EP3985111A1 (en) | 2015-08-19 | 2022-04-20 | Arc Bio, LLC | Capture of nucleic acids using a nucleic acid-guided nuclease-based system |
| CA2996001A1 (en) * | 2015-08-25 | 2017-03-02 | Duke University | Compositions and methods of improving specificity in genomic engineering using rna-guided endonucleases |
| CN108513584A (zh) | 2015-08-28 | 2018-09-07 | 先锋国际良种公司 | 苍白杆菌介导的植物转化 |
| US9512446B1 (en) | 2015-08-28 | 2016-12-06 | The General Hospital Corporation | Engineered CRISPR-Cas9 nucleases |
| CA2996888A1 (en) * | 2015-08-28 | 2017-03-09 | The General Hospital Corporation | Engineered crispr-cas9 nucleases |
| US9926546B2 (en) | 2015-08-28 | 2018-03-27 | The General Hospital Corporation | Engineered CRISPR-Cas9 nucleases |
| WO2017040511A1 (en) | 2015-08-31 | 2017-03-09 | Agilent Technologies, Inc. | Compounds and methods for crispr/cas-based genome editing by homologous recombination |
| US20170058272A1 (en) | 2015-08-31 | 2017-03-02 | Caribou Biosciences, Inc. | Directed nucleic acid repair |
| WO2017044419A1 (en) * | 2015-09-08 | 2017-03-16 | University Of Massachusetts | Dnase h activity of neisseria meningitidis cas9 |
| US11773455B2 (en) * | 2015-09-09 | 2023-10-03 | Psomagen, Inc. | Method and system for microbiome-derived diagnostics and therapeutics infectious disease and other health conditions associated with antibiotic usage |
| CN108271384B (zh) * | 2015-09-09 | 2022-04-15 | 国立大学法人神户大学 | 用于特异性转变靶向dna序列的核酸碱基的革兰氏阳性菌的基因组序列的转变方法、及其使用的分子复合体 |
| WO2017044885A1 (en) * | 2015-09-09 | 2017-03-16 | uBiome, Inc. | Method and system for microbiome-derived diagnostics and therapeutics for conditions associated with cerebro-craniofacial health |
| WO2017044776A1 (en) * | 2015-09-10 | 2017-03-16 | Texas Tech University System | Single-guide rna (sgrna) with improved knockout efficiency |
| CA3000816A1 (en) | 2015-09-11 | 2017-03-16 | The General Hospital Corporation | Full interrogation of nuclease dsbs and sequencing (find-seq) |
| US11247968B2 (en) | 2015-09-14 | 2022-02-15 | The Board Of Regents Of The University Of Texas System | Lipocationic dendrimers and uses thereof |
| EP3352795B1 (en) * | 2015-09-21 | 2020-08-12 | The Regents of The University of California | Compositions and methods for target nucleic acid modification |
| AU2016326711B2 (en) | 2015-09-24 | 2022-11-03 | Editas Medicine, Inc. | Use of exonucleases to improve CRISPR/Cas-mediated genome editing |
| WO2017053729A1 (en) | 2015-09-25 | 2017-03-30 | The Board Of Trustees Of The Leland Stanford Junior University | Nuclease-mediated genome editing of primary cells and enrichment thereof |
| WO2017058751A1 (en) * | 2015-09-28 | 2017-04-06 | North Carolina State University | Methods and compositions for sequence specific antimicrobials |
| WO2017058791A1 (en) * | 2015-09-29 | 2017-04-06 | Agenovir Corporation | Compositions and methods for treatment of latent viral infections |
| CA3000762A1 (en) | 2015-09-30 | 2017-04-06 | The General Hospital Corporation | Comprehensive in vitro reporting of cleavage events by sequencing (circle-seq) |
| WO2017059241A1 (en) | 2015-10-02 | 2017-04-06 | The United States Of America, As Represented By The Secretary, Department Of Health And Human Services | Lentiviral protein delivery system for rna-guided genome editing |
| WO2017062855A1 (en) | 2015-10-09 | 2017-04-13 | Monsanto Technology Llc | Novel rna-guided nucleases and uses thereof |
| WO2017066175A1 (en) | 2015-10-12 | 2017-04-20 | E. I. Du Pont De Nemours And Company | Protected dna templates for gene modification and increased homologous recombination in cells and methods of use |
| US11970710B2 (en) | 2015-10-13 | 2024-04-30 | Duke University | Genome engineering with Type I CRISPR systems in eukaryotic cells |
| US9862941B2 (en) | 2015-10-14 | 2018-01-09 | Pioneer Hi-Bred International, Inc. | Single cell microfluidic device |
| FR3042506B1 (fr) * | 2015-10-16 | 2018-11-30 | IFP Energies Nouvelles | Outil genetique de transformation de bacteries clostridium |
| EP4265633A3 (en) | 2015-10-16 | 2024-01-24 | The Trustees Of Columbia University In The City Of New York | Compositions and methods for inhibition of lineage specific antigens |
| JP7059179B2 (ja) | 2015-10-20 | 2022-04-25 | アンスティチュ ナショナル ドゥ ラ サンテ エ ドゥ ラ ルシェルシュ メディカル | 遺伝子操作のための方法及び製品 |
| MX2018004809A (es) | 2015-10-20 | 2018-06-08 | Pionner Hi Bred Int Inc | Metodos y composiciones para la modificacion genomica exenta de marcadores. |
| CA3024543A1 (en) | 2015-10-22 | 2017-04-27 | The Broad Institute, Inc. | Type vi-b crispr enzymes and systems |
| EP3365461B1 (en) | 2015-10-22 | 2020-10-14 | Institut National de la Sante et de la Recherche Medicale (INSERM) | Endonuclease-barcoding |
| US9677090B2 (en) | 2015-10-23 | 2017-06-13 | Caribou Biosciences, Inc. | Engineered nucleic-acid targeting nucleic acids |
| IL258821B (en) | 2015-10-23 | 2022-07-01 | Harvard College | Nucleobase editors and uses thereof |
| EP4279084B1 (en) | 2015-10-28 | 2025-06-11 | Vertex Pharmaceuticals Inc. | Materials and methods for treatment of duchenne muscular dystrophy |
| MX2018005611A (es) | 2015-11-03 | 2018-11-09 | Harvard College | Metodo y aparato para la formacion de imagenes volumetricas de una matriz tridimensional que contiene acido nucleico. |
| EP3371306B8 (en) | 2015-11-04 | 2023-02-22 | Vertex Pharmaceuticals Incorporated | Materials and methods for treatment of hemoglobinopathies |
| CA2999649A1 (en) | 2015-11-06 | 2017-05-11 | Crispr Therapeutics Ag | Materials and methods for treatment of glycogen storage disease type 1a |
| WO2017079724A1 (en) | 2015-11-06 | 2017-05-11 | The Jackson Laboratory | Large genomic dna knock-in and uses thereof |
| US20180258438A1 (en) | 2015-11-06 | 2018-09-13 | Pioneer Hi-Bred International, Inc. | Generation of complex trait loci in soybean and methods of use |
| US11566052B2 (en) | 2015-11-11 | 2023-01-31 | Lonza Ltd. | CRISPR-associated (Cas) proteins with reduced immunogenicity |
| CA2947904A1 (en) * | 2015-11-12 | 2017-05-12 | Pfizer Inc. | Tissue-specific genome engineering using crispr-cas9 |
| WO2017083766A1 (en) * | 2015-11-13 | 2017-05-18 | Massachusetts Institute Of Technology | High-throughput crispr-based library screening |
| EA201891192A1 (ru) | 2015-11-16 | 2019-01-31 | Рисёрч Инститьют Эт Нейшнвайд Чилдрен'С Хоспитал | Средства и способы лечения миопатий, связанных с титином, и других титинопатий |
| US10669320B2 (en) | 2015-11-18 | 2020-06-02 | The Regents Of The University Of Michigan | Mps1 and KNL1 phosphorylation system |
| JP6923205B2 (ja) | 2015-11-27 | 2021-08-18 | 国立大学法人神戸大学 | 標的化したdna配列の核酸塩基を特異的に変換する、単子葉植物のゲノム配列の変換方法、及びそれに用いる分子複合体 |
| US12239706B2 (en) | 2015-11-30 | 2025-03-04 | Seed Health, Inc. | Method and system for protecting monarch butterflies from pesticides |
| US10086024B2 (en) | 2015-11-30 | 2018-10-02 | Joseph E. Kovarik | Method and system for protecting honey bees, bats and butterflies from neonicotinoid pesticides |
| US10933128B2 (en) | 2015-11-30 | 2021-03-02 | Joseph E. Kovarik | Method and system for protecting honey bees from pesticides |
| JP7108307B2 (ja) | 2015-11-30 | 2022-07-28 | デューク ユニバーシティ | 遺伝子編集によるヒトジストロフィン遺伝子の修正用の治療標的および使用方法 |
| US11903974B2 (en) | 2015-11-30 | 2024-02-20 | Flagship Pioneering Innovations V, Inc. | Methods and compositions relating to chondrisomes from cultured cells |
| US11529412B2 (en) | 2015-11-30 | 2022-12-20 | Seed Health, Inc. | Method and system for protecting honey bees from pesticides |
| US10568916B2 (en) | 2015-11-30 | 2020-02-25 | Joseph E. Kovarik | Method and system for protecting honey bees, bats and butterflies from neonicotinoid pesticides |
| US10675347B2 (en) | 2015-11-30 | 2020-06-09 | Joseph E. Kovarik | Method and system for protecting honey bees from fipronil pesticides |
| US10946042B2 (en) | 2015-12-01 | 2021-03-16 | The Trustees Of The University Of Pennsylvania | Compositions and methods for selective phagocytosis of human cancer cells |
| CN109715801B (zh) | 2015-12-01 | 2022-11-01 | 克里斯普治疗股份公司 | 用于治疗α1抗胰蛋白酶缺乏的材料和方法 |
| US9988624B2 (en) | 2015-12-07 | 2018-06-05 | Zymergen Inc. | Microbial strain improvement by a HTP genomic engineering platform |
| KR20180084756A (ko) | 2015-12-07 | 2018-07-25 | 지머젠 인코포레이티드 | 코리네박테리움 글루타미컴으로부터의 프로모터 |
| DK3386550T3 (da) | 2015-12-07 | 2021-04-26 | Arc Bio Llc | Fremgangsmåder til fremstilling og anvendelse af guide nukleinsyrer |
| US11208649B2 (en) | 2015-12-07 | 2021-12-28 | Zymergen Inc. | HTP genomic engineering platform |
| US20180362961A1 (en) | 2015-12-11 | 2018-12-20 | Danisco Us Inc. | Methods and compositions for enhanced nuclease-mediated genome modification and reduced off-target site effects |
| US20190075770A1 (en) * | 2015-12-18 | 2019-03-14 | Japan Science And Technology Agency | Genetic modification non-human organism, egg cells, fertilized eggs, and method for modifying target genes |
| DK3390631T3 (da) | 2015-12-18 | 2020-07-13 | Danisco Us Inc | Fremgangsmåder og sammensætninger til t-rna-baseret guide-rna-ekspression |
| US11118194B2 (en) | 2015-12-18 | 2021-09-14 | The Regents Of The University Of California | Modified site-directed modifying polypeptides and methods of use thereof |
| WO2017106767A1 (en) * | 2015-12-18 | 2017-06-22 | The Scripps Research Institute | Production of unnatural nucleotides using a crispr/cas9 system |
| KR102698595B1 (ko) * | 2015-12-18 | 2024-08-23 | 다니스코 유에스 인크. | 중합효소 ii(pol-ii) 기반의 가이드 rna 발현을 위한 방법 및 조성물 |
| WO2017106795A1 (en) | 2015-12-18 | 2017-06-22 | Oncosec Medical Incorporated | Plasmid constructs for heterologous protein expression and methods of use |
| US11542466B2 (en) | 2015-12-22 | 2023-01-03 | North Carolina State University | Methods and compositions for delivery of CRISPR based antimicrobials |
| EP3394092A1 (en) | 2015-12-23 | 2018-10-31 | Fred Hutchinson Cancer Research Center | High affinity t cell receptors and uses thereof |
| MX2018007840A (es) | 2015-12-23 | 2019-05-02 | Crispr Therapeutics Ag | Materiales y metodos para el tratamiento de la esclerosis lateral amiotrofica y/o la degeneracion lobar frontotemporal. |
| EP3397756B1 (en) | 2015-12-30 | 2023-03-08 | Novartis AG | Immune effector cell therapies with enhanced efficacy |
| BR112018013663A2 (pt) | 2016-01-11 | 2019-01-22 | Univ Leland Stanford Junior | proteínas quiméricas e métodos de imunoterapia |
| US11441146B2 (en) | 2016-01-11 | 2022-09-13 | Christiana Care Health Services, Inc. | Compositions and methods for improving homogeneity of DNA generated using a CRISPR/Cas9 cleavage system |
| KR20180096800A (ko) | 2016-01-11 | 2018-08-29 | 더 보드 어브 트러스티스 어브 더 리랜드 스탠포드 주니어 유니버시티 | 키메라 단백질 및 유전자 발현을 조절하는 방법 |
| US11427837B2 (en) * | 2016-01-12 | 2022-08-30 | The Regents Of The University Of California | Compositions and methods for enhanced genome editing |
| AU2017208013B2 (en) | 2016-01-15 | 2022-12-01 | Beth Israel Deaconess Medical Center, Inc. | Therapeutic use of mitochondria and combined mitochondrial agents |
| WO2017124086A1 (en) | 2016-01-15 | 2017-07-20 | The Jackson Laboratory | Genetically modified non-human mammals by multi-cycle electroporation of cas9 protein |
| US11136589B2 (en) | 2016-01-26 | 2021-10-05 | Pioneer Hi-Bred International, Inc. | Waxy corn |
| EP3199632A1 (en) | 2016-01-26 | 2017-08-02 | ACIB GmbH | Temperature-inducible crispr/cas system |
| US11518994B2 (en) * | 2016-01-30 | 2022-12-06 | Bonac Corporation | Artificial single guide RNA and use thereof |
| US20190038771A1 (en) | 2016-02-02 | 2019-02-07 | Crispr Therapeutics Ag | Materials and methods for treatment of severe combined immunodeficiency (scid) or omenn syndrome |
| US11845933B2 (en) | 2016-02-03 | 2023-12-19 | Massachusetts Institute Of Technology | Structure-guided chemical modification of guide RNA and its applications |
| US11542515B2 (en) | 2016-02-09 | 2023-01-03 | Cibus Us Llc | Methods and compositions for increasing efficiency of targeted gene modification using oligonucleotide-mediated gene repair |
| US20190048415A1 (en) | 2016-02-10 | 2019-02-14 | The Regents Of The University Of Michigan | Detection of nucleic acids |
| US11339427B2 (en) | 2016-02-12 | 2022-05-24 | Jumpcode Genomics, Inc. | Method for target specific RNA transcription of DNA sequences |
| WO2017139309A1 (en) | 2016-02-12 | 2017-08-17 | Ceres, Inc. | Methods and materials for high throughput testing of mutagenized allele combinations |
| US9896696B2 (en) | 2016-02-15 | 2018-02-20 | Benson Hill Biosystems, Inc. | Compositions and methods for modifying genomes |
| AU2017221424A1 (en) | 2016-02-16 | 2018-09-20 | Yale University | Compositions and methods for treatment of cystic fibrosis |
| CA3014792A1 (en) | 2016-02-16 | 2017-08-24 | Carnegie Mellon University | Compositions for enhancing targeted gene editing and methods of use thereof |
| EP3416689B1 (en) | 2016-02-18 | 2023-01-18 | CRISPR Therapeutics AG | Materials and methods for treatment of severe combined immunodeficiency (scid) or omenn syndrome |
| US20190249172A1 (en) | 2016-02-18 | 2019-08-15 | The Regents Of The University Of California | Methods and compositions for gene editing in stem cells |
| US11155814B2 (en) | 2016-02-22 | 2021-10-26 | Caribou Biosciences, Inc. | Methods for using DNA repair for cell engineering |
| CN105646719B (zh) * | 2016-02-24 | 2019-12-20 | 无锡市妇幼保健院 | 一种高效定点转基因的工具及其应用 |
| WO2017147278A1 (en) | 2016-02-25 | 2017-08-31 | The Children's Medical Center Corporation | Customized class switch of immunoglobulin genes in lymphoma and hybridoma by crispr/cas9 technology |
| CN109072245B (zh) * | 2016-02-26 | 2022-06-14 | 朗泽科技新西兰有限公司 | 用于c1固定菌的crispr/cas系统 |
| WO2017151444A1 (en) | 2016-02-29 | 2017-09-08 | Agilent Technologies, Inc. | Methods and compositions for blocking off-target nucleic acids from cleavage by crispr proteins |
| JP2019507610A (ja) | 2016-03-04 | 2019-03-22 | インドア バイオテクノロジーズ インコーポレイテッド | CRISPR−Casゲノム編集に基づく、Fel d1ノックアウト並びに関連組成物及び方法 |
| WO2017155714A1 (en) | 2016-03-11 | 2017-09-14 | Pioneer Hi-Bred International, Inc. | Novel cas9 systems and methods of use |
| WO2017155715A1 (en) | 2016-03-11 | 2017-09-14 | Pioneer Hi-Bred International, Inc. | Novel cas9 systems and methods of use |
| EP3699280A3 (en) | 2016-03-11 | 2020-11-18 | Pioneer Hi-Bred International, Inc. | Novel cas9 systems and methods of use |
| CN109152848B (zh) * | 2016-03-15 | 2022-12-09 | 马萨诸塞大学 | 抗-crispr化合物以及使用方法 |
| CN109414414A (zh) | 2016-03-16 | 2019-03-01 | 戴维·格拉德斯通研究所 | 用于治疗肥胖症和/或糖尿病以及用于鉴定候选治疗剂的方法和组合物 |
| US11083799B2 (en) | 2016-03-16 | 2021-08-10 | Crispr Therapeutics Ag | Materials and methods for treatment of hereditary haemochromatosis |
| EP3219799A1 (en) | 2016-03-17 | 2017-09-20 | IMBA-Institut für Molekulare Biotechnologie GmbH | Conditional crispr sgrna expression |
| EP3433363A1 (en) | 2016-03-25 | 2019-01-30 | Editas Medicine, Inc. | Genome editing systems comprising repair-modulating enzyme molecules and methods of their use |
| US11512311B2 (en) | 2016-03-25 | 2022-11-29 | Editas Medicine, Inc. | Systems and methods for treating alpha 1-antitrypsin (A1AT) deficiency |
| WO2017172860A1 (en) * | 2016-03-31 | 2017-10-05 | President And Fellows Of Harvard College | Methods and compositions for the single tube preparation of sequencing libraries using cas9 |
| US20190117799A1 (en) | 2016-04-01 | 2019-04-25 | The Brigham And Women's Hospital, Inc. | Stimuli-responsive nanoparticles for biomedical applications |
| EP4047092A1 (en) | 2016-04-13 | 2022-08-24 | Editas Medicine, Inc. | Cas9 fusion molecules, gene editing systems, and methods of use thereof |
| EP3443081A4 (en) * | 2016-04-13 | 2019-10-30 | Duke University | CRISPR / CAS9-BASED REPRESSORS FOR IN VIVO SHUT-OFF OF GEN-TARGETS AND METHOD OF USE |
| CN109312308A (zh) * | 2016-04-14 | 2019-02-05 | 亿阳集团美国硅谷公司 | 通过使用核酸酶来进行人神经干细胞的基因组编辑 |
| EP3443096B1 (en) | 2016-04-15 | 2023-03-01 | Novartis AG | Compositions and methods for selective expression of chimeric antigen receptors |
| SG11201808831TA (en) | 2016-04-15 | 2018-11-29 | Memorial Sloan Kettering Cancer Center | Transgenic t cell and chimeric antigen receptor t cell compositions and related methods |
| WO2017181107A2 (en) * | 2016-04-16 | 2017-10-19 | Ohio State Innovation Foundation | Modified cpf1 mrna, modified guide rna, and uses thereof |
| US20200330609A1 (en) | 2016-04-18 | 2020-10-22 | Crispr Therapeutics Ag | Materials and methods for treatment of hemoglobinopathies |
| MX2018012873A (es) * | 2016-04-22 | 2019-08-05 | Intellia Therapeutics Inc | Composiciones y métodos para el tratar enfermedades asociadas con repeticiones de trinucleótidos en el factor de transcripción cuatro. |
| EP3449016B1 (en) | 2016-04-25 | 2025-07-23 | President and Fellows of Harvard College | Hybridization chain reaction methods for in situ molecular detection |
| EP3448996A1 (en) | 2016-04-26 | 2019-03-06 | Massachusetts Institute of Technology | Extensible recombinase cascades |
| EP3448997B1 (en) | 2016-04-27 | 2020-10-14 | Massachusetts Institute of Technology | Stable nanoscale nucleic acid assemblies and methods thereof |
| US11514331B2 (en) | 2016-04-27 | 2022-11-29 | Massachusetts Institute Of Technology | Sequence-controlled polymer random access memory storage |
| WO2017191503A1 (en) | 2016-05-05 | 2017-11-09 | Crispr Therapeutics Ag | Materials and methods for treatment of hemoglobinopathies |
| JP7169880B2 (ja) | 2016-05-06 | 2022-11-11 | ザ ブリガム アンド ウィメンズ ホスピタル インコーポレイテッド | 軟骨へのカプセル化薬剤の制御送達のための二成分型自己集合ゲル |
| US20190300872A1 (en) * | 2016-05-06 | 2019-10-03 | Tod M. Woolf | Improved Methods of Genome Editing with and without Programmable Nucleases |
| WO2017197128A1 (en) | 2016-05-11 | 2017-11-16 | Yale University | Poly(amine-co-ester) nanoparticles and methods of use thereof |
| EP3456181A4 (en) | 2016-05-13 | 2019-12-25 | Kaneka Corporation | METHOD FOR PRODUCING A TRANSFORMED PLANT |
| US11499158B2 (en) | 2016-05-13 | 2022-11-15 | Kaneka Corporation | Method for modifying plant |
| EP3456825A4 (en) * | 2016-05-13 | 2020-03-18 | Kaneka Corporation | METHOD FOR PLANT GENE EDITING |
| EP4368637A3 (en) | 2016-05-20 | 2024-07-10 | Regeneron Pharmaceuticals, Inc. | Methods for breaking immunological tolerance using multiple guide rnas |
| CN109689677B (zh) * | 2016-05-27 | 2023-11-10 | 阿迪根有限公司 | 用于基因组编辑分子的细胞内递送的肽和纳米颗粒 |
| EP3463484A4 (en) | 2016-05-27 | 2019-10-30 | The Regents of the University of California | METHOD AND COMPOSITIONS FOR TARGETING RNA POLYMERASES AND NON-CODING RNA BIOGENESIS AT SPECIFIC LOCI |
| LT3604527T (lt) | 2016-06-02 | 2021-06-25 | Sigma-Aldrich Co., Llc | Programuojamų, dnr surišančių baltymų panaudojimas tiksliniam genomo modifikavimui pagerinti |
| GB201609811D0 (en) | 2016-06-05 | 2016-07-20 | Snipr Technologies Ltd | Methods, cells, systems, arrays, RNA and kits |
| US10767175B2 (en) | 2016-06-08 | 2020-09-08 | Agilent Technologies, Inc. | High specificity genome editing using chemically modified guide RNAs |
| WO2017222834A1 (en) * | 2016-06-10 | 2017-12-28 | City Of Hope | Compositions and methods for mitochondrial genome editing |
| AU2017286122A1 (en) | 2016-06-14 | 2018-11-22 | Pioneer Hi-Bred International, Inc. | Use of Cpf1 endonuclease for plant genome modifications |
| CN109312386B (zh) * | 2016-06-15 | 2022-10-25 | 株式会社图尔金 | 使用中靶靶标和脱靶靶标的多重靶标系统筛选靶特异性核酸酶的方法及其用途 |
| US10337051B2 (en) | 2016-06-16 | 2019-07-02 | The Regents Of The University Of California | Methods and compositions for detecting a target RNA |
| KR20190019168A (ko) * | 2016-06-17 | 2019-02-26 | 더 브로드 인스티튜트, 인코퍼레이티드 | 제vi형 crispr 오솔로그 및 시스템 |
| EP3472310A1 (en) | 2016-06-20 | 2019-04-24 | Pioneer Hi-Bred International, Inc. | Novel cas systems and methods of use |
| US11293021B1 (en) | 2016-06-23 | 2022-04-05 | Inscripta, Inc. | Automated cell processing methods, modules, instruments, and systems |
| CN109688820B (zh) | 2016-06-24 | 2023-01-10 | 科罗拉多州立大学董事会(法人团体) | 用于生成条形码化组合文库的方法 |
| EP3474849B1 (en) | 2016-06-27 | 2025-05-21 | The Broad Institute, Inc. | Compositions and methods for detecting and treating diabetes |
| WO2018005589A1 (en) | 2016-06-28 | 2018-01-04 | Cellectis | Altering expression of gene products in plants through targeted insertion of nucleic acid sequences |
| WO2018002762A1 (en) | 2016-06-29 | 2018-01-04 | Crispr Therapeutics Ag | Materials and methods for treatment of amyotrophic lateral sclerosis (als) and other related disorders |
| US11427838B2 (en) | 2016-06-29 | 2022-08-30 | Vertex Pharmaceuticals Incorporated | Materials and methods for treatment of myotonic dystrophy type 1 (DM1) and other related disorders |
| AU2017290614C1 (en) | 2016-06-29 | 2024-01-18 | Crispr Therapeutics Ag | Materials and methods for treatment of friedreich ataxia and other related disorders |
| KR102345899B1 (ko) | 2016-06-30 | 2021-12-31 | 지머젠 인코포레이티드 | 박테리아 헤모글로빈 라이브러리를 생성하는 방법 및 이의 용도 |
| EP3478845A4 (en) | 2016-06-30 | 2019-07-31 | Zymergen, Inc. | METHOD OF GENERATING A GLUCOSE PERMEASE LIBRARY AND USES THEREOF |
| AU2017292169B2 (en) | 2016-07-06 | 2021-12-23 | Vertex Pharmaceuticals Incorporated | Materials and methods for treatment of pain related disorders |
| US11459587B2 (en) | 2016-07-06 | 2022-10-04 | Vertex Pharmaceuticals Incorporated | Materials and methods for treatment of pain related disorders |
| WO2018007871A1 (en) | 2016-07-08 | 2018-01-11 | Crispr Therapeutics Ag | Materials and methods for treatment of transthyretin amyloidosis |
| WO2018013932A1 (en) | 2016-07-15 | 2018-01-18 | Salk Institute For Biological Studies | Methods and compositions for genome editing in non-dividing cells |
| CN120285179A (zh) | 2016-07-15 | 2025-07-11 | 诺华股份有限公司 | 使用与激酶抑制剂组合的嵌合抗原受体治疗和预防细胞因子释放综合征 |
| JP7490211B2 (ja) | 2016-07-19 | 2024-05-27 | デューク ユニバーシティ | Cpf1に基づくゲノム編集の治療適用 |
| WO2018015444A1 (en) | 2016-07-22 | 2018-01-25 | Novozymes A/S | Crispr-cas9 genome editing with multiple guide rnas in filamentous fungi |
| WO2018020323A2 (en) | 2016-07-25 | 2018-02-01 | Crispr Therapeutics Ag | Materials and methods for treatment of fatty acid disorders |
| CN106191043B (zh) * | 2016-07-26 | 2019-07-02 | 吉林大学 | 一种基因片段、载体pPlasmid-Clearance及应用 |
| EP4230648A3 (en) | 2016-07-28 | 2023-10-18 | Regeneron Pharmaceuticals, Inc. | Gpr156 variants and uses thereof |
| KR20190041476A (ko) | 2016-07-29 | 2019-04-22 | 리제너론 파마슈티칼스 인코포레이티드 | C-절단된 피브릴린-1의 발현을 유도하는 돌연변이를 포함하는 마우스 |
| BR112019001887A2 (pt) | 2016-08-02 | 2019-07-09 | Editas Medicine Inc | composições e métodos para o tratamento de doença associada a cep290 |
| JP7231935B2 (ja) | 2016-08-03 | 2023-03-08 | プレジデント アンド フェローズ オブ ハーバード カレッジ | アデノシン核酸塩基編集因子およびそれらの使用 |
| US11078481B1 (en) | 2016-08-03 | 2021-08-03 | KSQ Therapeutics, Inc. | Methods for screening for cancer targets |
| JOP20170161A1 (ar) | 2016-08-04 | 2019-01-30 | Arrowhead Pharmaceuticals Inc | عوامل RNAi للعدوى بفيروس التهاب الكبد ب |
| CA3033327A1 (en) | 2016-08-09 | 2018-02-15 | President And Fellows Of Harvard College | Programmable cas9-recombinase fusion proteins and uses thereof |
| KR101710026B1 (ko) | 2016-08-10 | 2017-02-27 | 주식회사 무진메디 | Cas9 단백질 및 가이드 RNA의 혼성체를 함유하는 나노 리포좀 전달체 조성물 |
| WO2018031950A1 (en) | 2016-08-12 | 2018-02-15 | Caribou Biosciences, Inc. | Protein engineering methods |
| EP3500669A4 (en) * | 2016-08-16 | 2020-01-22 | The Regents of the University of California | METHOD FOR SEARCHING FOR LOW ABUNDANCE SEQUENCES BY HYBRIDIZATION (FLASH) |
| MA46018A (fr) * | 2016-08-19 | 2019-06-26 | Bluebird Bio Inc | Activateurs d'édition du génome |
| US11434476B2 (en) * | 2016-08-19 | 2022-09-06 | Whitehead Institute For Biomedical Research | Methods of editing DNA methylation |
| EP3500677A4 (en) * | 2016-08-20 | 2020-04-01 | Avellino Lab USA, Inc. | Single guide rna, crispr/cas9 systems, and methods of use thereof |
| WO2018039438A1 (en) | 2016-08-24 | 2018-03-01 | President And Fellows Of Harvard College | Incorporation of unnatural amino acids into proteins using base editing |
| CA3035414C (en) | 2016-08-29 | 2022-02-22 | The Regents Of The University Of California | Topical formulations based on ionic species for skin treatment |
| US11078483B1 (en) | 2016-09-02 | 2021-08-03 | KSQ Therapeutics, Inc. | Methods for measuring and improving CRISPR reagent function |
| CN106399311A (zh) * | 2016-09-07 | 2017-02-15 | 同济大学 | 用于Chip‑seq全基因组结合谱的内源蛋白标记的方法 |
| US20190242908A1 (en) | 2016-09-08 | 2019-08-08 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods for diagnosing and treating nephrotic syndrome |
| EP4431607A3 (en) | 2016-09-09 | 2024-12-11 | The Board of Trustees of the Leland Stanford Junior University | High-throughput precision genome editing |
| AU2017327384B2 (en) | 2016-09-13 | 2021-05-20 | The Jackson Laboratory | Targeted enhanced DNA demethylation |
| MX2019003176A (es) | 2016-09-23 | 2019-07-04 | Hutchinson Fred Cancer Res | Receptores de linfocitos t (tcr) especificos del antigeno menor de histocompatibilidad (h) ah-1 y sus usos. |
| US20190275171A1 (en) * | 2016-09-23 | 2019-09-12 | Ionis Pharmaceuticals, Inc. | Gene therapy and targeted delivery of conjugated compounds |
| US20190225974A1 (en) | 2016-09-23 | 2019-07-25 | BASF Agricultural Solutions Seed US LLC | Targeted genome optimization in plants |
| US20190203230A1 (en) | 2016-09-28 | 2019-07-04 | Novartis Ag | Porous membrane-based macromolecule delivery system |
| GB201616590D0 (en) | 2016-09-29 | 2016-11-16 | Oxford Nanopore Technologies Limited | Method |
| WO2018060238A1 (en) * | 2016-09-29 | 2018-04-05 | F. Hoffmann-La Roche Ag | Method to analyze and optimize gene editing modules and delivery approaches |
| EP3523426A4 (en) * | 2016-09-30 | 2020-01-22 | The Regents of The University of California | RNA GUIDED NUCLEIC ACID MODIFYING ENZYMES AND METHOD FOR USE THEREOF |
| GB2569734B (en) * | 2016-09-30 | 2022-09-07 | Univ California | RNA-guided nucleic acid modifying enzymes and methods of use thereof |
| US10669539B2 (en) | 2016-10-06 | 2020-06-02 | Pioneer Biolabs, Llc | Methods and compositions for generating CRISPR guide RNA libraries |
| KR102717188B1 (ko) | 2016-10-07 | 2024-10-16 | 노파르티스 아게 | 암의 치료를 위한 키메라 항원 수용체 |
| WO2018069232A1 (en) | 2016-10-10 | 2018-04-19 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods for predicting the risk of having cardiac hypertrophy |
| US11090653B2 (en) | 2016-10-11 | 2021-08-17 | The Regents Of The University Of California | Systems and methods to encapsulate and preserve organic matter for analysis |
| EP3525578A1 (en) | 2016-10-13 | 2019-08-21 | Pioneer Hi-Bred International, Inc. | Generating northern leaf blight resistant maize |
| AU2017342543B2 (en) | 2016-10-14 | 2024-06-27 | President And Fellows Of Harvard College | AAV delivery of nucleobase editors |
| KR20190067209A (ko) | 2016-10-14 | 2019-06-14 | 더 제너럴 하스피탈 코포레이션 | 후성적으로 조절되는 부위-특이적 뉴클레아제 |
| GB201617559D0 (en) | 2016-10-17 | 2016-11-30 | University Court Of The University Of Edinburgh The | Swine comprising modified cd163 and associated methods |
| EP3529359B1 (en) | 2016-10-18 | 2023-12-13 | Regents of the University of Minnesota | Tumor infiltrating lymphocytes for use in therapy |
| US11766400B2 (en) | 2016-10-24 | 2023-09-26 | Yale University | Biodegradable contraceptive implants |
| US20180119141A1 (en) | 2016-10-28 | 2018-05-03 | Massachusetts Institute Of Technology | Crispr/cas global regulator screening platform |
| WO2018081531A2 (en) | 2016-10-28 | 2018-05-03 | Ariad Pharmaceuticals, Inc. | Methods for human t-cell activation |
| EP3535396A1 (en) | 2016-11-01 | 2019-09-11 | Novartis AG | Methods and compositions for enhancing gene editing |
| US11732258B2 (en) * | 2016-11-02 | 2023-08-22 | President And Fellows Of Harvard College | Engineered guide RNA sequences for in situ detection and sequencing |
| WO2018081978A1 (zh) * | 2016-11-03 | 2018-05-11 | 深圳华大基因研究院 | 提高基因编辑效率的方法和系统 |
| EP3535416A4 (en) | 2016-11-04 | 2020-05-13 | Flagship Pioneering Innovations V. Inc. | NEW PLANT CELLS, PLANTS AND SEEDS |
| CN109906030B (zh) | 2016-11-04 | 2022-03-18 | 安健基因公司 | 用于产生仅重链抗体的经基因修饰的非人动物和方法 |
| US20190345500A1 (en) | 2016-11-14 | 2019-11-14 | |Nserm (Institut National De La Santé Et De La Recherche Médicale) | Methods and pharmaceutical compositions for modulating stem cells proliferation or differentiation |
| WO2018094356A2 (en) * | 2016-11-18 | 2018-05-24 | Genedit Inc. | Compositions and methods for target nucleic acid modification |
| CN118562882A (zh) | 2016-11-22 | 2024-08-30 | 新加坡国立大学 | 用于t细胞恶性肿瘤免疫疗法的cd7表达阻滞剂和嵌合抗原受体 |
| WO2018098480A1 (en) | 2016-11-28 | 2018-05-31 | The Board Of Regents Of The University Of Texas System | Prevention of muscular dystrophy by crispr/cpf1-mediated gene editing |
| US20200081010A1 (en) | 2016-12-02 | 2020-03-12 | Inserm (Institut National De La Sante Et De La Recherche Medicale) | Methods and compositions for diagnosing renal cell carcinoma |
| US9816093B1 (en) | 2016-12-06 | 2017-11-14 | Caribou Biosciences, Inc. | Engineered nucleic acid-targeting nucleic acids |
| JP2020500541A (ja) | 2016-12-08 | 2020-01-16 | ザ ボード オブ リージェンツ オブ ザ ユニバーシティー オブ テキサス システム | ヒト化デュシェンヌ型筋ジストロフィー変異を有するdmdレポーターモデル |
| US20200149039A1 (en) | 2016-12-12 | 2020-05-14 | Whitehead Institute For Biomedical Research | Regulation of transcription through ctcf loop anchors |
| CN110191955B (zh) | 2016-12-13 | 2024-05-31 | 西雅图儿童医院(Dba西雅图儿童研究所) | 在体外和体内对工程化的细胞中表达的化学物质诱导的信号传导复合物进行外源性药物激活的方法 |
| EP3555275A1 (en) | 2016-12-14 | 2019-10-23 | Wageningen Universiteit | Thermostable cas9 nucleases |
| WO2018112278A1 (en) | 2016-12-14 | 2018-06-21 | Ligandal, Inc. | Methods and compositions for nucleic acid and protein payload delivery |
| US20200085758A1 (en) | 2016-12-16 | 2020-03-19 | The Brigham And Women's Hospital, Inc. | Co-delivery of nucleic acids for simultaneous suppression and expression of target genes |
| EP3555297A1 (en) | 2016-12-19 | 2019-10-23 | Editas Medicine, Inc. | Assessing nuclease cleavage |
| WO2018118585A1 (en) * | 2016-12-22 | 2018-06-28 | Agenovir Corporation | Antiviral compositions |
| MX2019007546A (es) | 2016-12-22 | 2019-10-24 | Monsanto Technology Llc | Modificación genética de cultivos a base de edición del genómica y producción de plantas braquíticas. |
| US20180237787A1 (en) * | 2016-12-23 | 2018-08-23 | President And Fellows Of Harvard College | Gene editing of pcsk9 |
| US10745677B2 (en) | 2016-12-23 | 2020-08-18 | President And Fellows Of Harvard College | Editing of CCR5 receptor gene to protect against HIV infection |
| WO2018126087A1 (en) | 2016-12-29 | 2018-07-05 | Applied Stemcell, Inc. | Gene editing method using virus |
| EP3342868B1 (en) | 2016-12-30 | 2019-12-25 | Systasy Bioscience GmbH | Constructs and screening methods |
| US11859219B1 (en) | 2016-12-30 | 2024-01-02 | Flagship Pioneering Innovations V, Inc. | Methods of altering a target nucleotide sequence with an RNA-guided nuclease and a single guide RNA |
| JOP20190166A1 (ar) | 2017-01-05 | 2019-07-02 | Univ Texas | استراتيجية مثلى من أجل تعديلات تخطي إكسون باستخدام crispr/cas9 مع متواليات توجيه ثلاثي |
| US12065666B2 (en) | 2017-01-05 | 2024-08-20 | Rutgers, The State University Of New Jersey | Targeted gene editing platform independent of DNA double strand break and uses thereof |
| WO2018127585A1 (en) | 2017-01-06 | 2018-07-12 | Txcell | Monospecific regulatory t cell population with cytotoxicity for b cells |
| EP3565907B1 (en) | 2017-01-06 | 2022-05-04 | Editas Medicine, Inc. | Methods of assessing nuclease cleavage |
| EP3346001A1 (en) | 2017-01-06 | 2018-07-11 | TXCell | Monospecific regulatory t cell population with cytotoxicity for b cells |
| JP7348064B2 (ja) | 2017-01-09 | 2023-09-20 | アポセンス リミテッド | 分子を経膜送達するための化合物及び方法 |
| WO2018129544A1 (en) | 2017-01-09 | 2018-07-12 | Whitehead Institute For Biomedical Research | Methods of altering gene expression by perturbing transcription factor multimers that structure regulatory loops |
| EP3568470B1 (en) | 2017-01-10 | 2022-07-06 | Christiana Care Health Services, Inc. | Methods for in vitro site-directed mutagenesis using gene editing technologies |
| EP3569708A4 (en) * | 2017-01-13 | 2020-09-09 | Jichi Medical University | AAV VECTOR FOR THE DISRUPTION OF THE COAGULATION-RELATED FACTOR GENE ON THE LIVER GENOME |
| CA3047416A1 (en) | 2017-01-20 | 2018-07-26 | The Regents Of The University Of California | Targeted gene activation in plants |
| RU2760851C2 (ru) | 2017-01-23 | 2021-11-30 | Регенерон Фармасьютикалз, Инк. | Варианты hsd17b13 и их применения |
| EP3574005B1 (en) | 2017-01-26 | 2021-12-15 | Novartis AG | Cd28 compositions and methods for chimeric antigen receptor therapy |
| EP3574102A4 (en) | 2017-01-26 | 2020-09-30 | The Regents of The University of California | TARGETED DEMETHYLATION OF GENES IN PLANTS |
| AU2018212986B2 (en) | 2017-01-28 | 2024-07-25 | Inari Agriculture Technology, Inc. | Novel plant cells, plants, and seeds |
| CN110582509A (zh) | 2017-01-31 | 2019-12-17 | 诺华股份有限公司 | 使用具有多特异性的嵌合t细胞受体蛋白治疗癌症 |
| US11345938B2 (en) | 2017-02-02 | 2022-05-31 | Cargill, Incorporated | Genetically modified cells that produce C6-C10 fatty acid derivatives |
| TW201839136A (zh) | 2017-02-06 | 2018-11-01 | 瑞士商諾華公司 | 治療血色素異常症之組合物及方法 |
| WO2018203947A2 (en) | 2017-02-06 | 2018-11-08 | Zymergen Inc. | Engineered biosynthetic pathways for production of tyramine by fermentation |
| EP3580212A4 (en) | 2017-02-08 | 2021-03-17 | Dana Farber Cancer Institute, Inc. | REGULATION OF CHEMERIC ANTIGEN RECEPTORS |
| JP7341060B2 (ja) | 2017-02-10 | 2023-09-08 | アンスティチュ ナショナル ドゥ ラ サンテ エ ドゥ ラ ルシェルシュ メディカル | Mapk経路の活性化に関連付けられる癌の処置のための方法及び医薬組成物 |
| JP2020507312A (ja) | 2017-02-10 | 2020-03-12 | ザイマージェン インコーポレイテッド | 複数の宿主用の複数のdnaコンストラクトのアセンブリ及び編集のためのモジュラーユニバーサルプラスミド設計戦略 |
| CN118256413A (zh) | 2017-02-21 | 2024-06-28 | 杜克大学 | 用于稳健动态代谢控制的组合物和方法 |
| EP3585900B1 (en) | 2017-02-22 | 2022-12-21 | CRISPR Therapeutics AG | Materials and methods for treatment of spinocerebellar ataxia type 2 (sca2) and other spinocerebellar ataxia type 2 protein (atxn2) gene related conditions or disorders |
| US11559588B2 (en) | 2017-02-22 | 2023-01-24 | Crispr Therapeutics Ag | Materials and methods for treatment of Spinocerebellar Ataxia Type 1 (SCA1) and other Spinocerebellar Ataxia Type 1 Protein (ATXN1) gene related conditions or disorders |
| CA3054031A1 (en) | 2017-02-22 | 2018-08-30 | Crispr Therapeutics Ag | Compositions and methods for gene editing |
| US11407997B2 (en) | 2017-02-22 | 2022-08-09 | Crispr Therapeutics Ag | Materials and methods for treatment of primary hyperoxaluria type 1 (PH1) and other alanine-glyoxylate aminotransferase (AGXT) gene related conditions or disorders |
| EP3585807A1 (en) | 2017-02-22 | 2020-01-01 | CRISPR Therapeutics AG | Materials and methods for treatment of early onset parkinson's disease (park1) and other synuclein, alpha (snca) gene related conditions or disorders |
| WO2018160731A1 (en) | 2017-02-28 | 2018-09-07 | Novartis Ag | Shp inhibitor compositions and uses for chimeric antigen receptor therapy |
| SG11201907870VA (en) | 2017-02-28 | 2019-09-27 | Vor Biopharma Inc | Compositions and methods for inhibition of lineage specific proteins |
| EP3592853A1 (en) | 2017-03-09 | 2020-01-15 | President and Fellows of Harvard College | Suppression of pain by gene editing |
| WO2018165629A1 (en) | 2017-03-10 | 2018-09-13 | President And Fellows Of Harvard College | Cytosine to guanine base editor |
| WO2018170184A1 (en) | 2017-03-14 | 2018-09-20 | Editas Medicine, Inc. | Systems and methods for the treatment of hemoglobinopathies |
| JP2020513814A (ja) * | 2017-03-14 | 2020-05-21 | ザ リージェンツ オブ ザ ユニバーシティ オブ カリフォルニア | Crispr cas9免疫ステルス(immune stealth)の操作 |
| EP3595708A4 (en) | 2017-03-15 | 2020-12-16 | Fred Hutchinson Cancer Research Center | HIGH AFFINITY MAGE-A1 SPECIFIC TCRS AND THEIR USES |
| WO2018167119A1 (en) | 2017-03-15 | 2018-09-20 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Pharmaceutical compositions for the treatment of thrombosis in patients suffering from a myeloproliferative neoplasm |
| GB2575930A (en) | 2017-03-23 | 2020-01-29 | Harvard College | Nucleobase editors comprising nucleic acid programmable DNA binding proteins |
| WO2018172570A1 (en) | 2017-03-24 | 2018-09-27 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Gfi1 inhibitors for the treatment of hyperglycemia |
| WO2018172508A1 (en) | 2017-03-24 | 2018-09-27 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods and compositions for treating melanoma |
| AU2018240515B2 (en) * | 2017-03-24 | 2024-07-25 | CureVac SE | Nucleic acids encoding CRISPR-associated proteins and uses thereof |
| PT3526324T (pt) | 2017-03-28 | 2021-10-20 | Locanabio Inc | Proteína associada a crispr (cas) |
| JP2020515588A (ja) | 2017-03-30 | 2020-05-28 | アンスティチュ ナショナル ドゥ ラ サンテ エ ドゥ ラ ルシェルシュ メディカル | ミトコンドリア遺伝子疾患の処置方法 |
| US11661624B2 (en) | 2017-03-30 | 2023-05-30 | Pioneer Hi-Bred International, Inc. | Methods of identifying and characterizing gene editing variations in nucleic acids |
| WO2018187493A1 (en) | 2017-04-04 | 2018-10-11 | Yale University | Compositions and methods for in utero delivery |
| AU2018250727A1 (en) | 2017-04-11 | 2019-10-31 | Regeneron Pharmaceuticals, Inc. | Assays for screening activity of modulators of members of the hydroxysteroid (17-beta) dehydrogenase (HSD17B) family |
| JP7248586B2 (ja) | 2017-04-14 | 2023-03-29 | チルドレンズ ホスピタル メディカル センター | 複数ドナー幹細胞組成物およびそれを作製する方法 |
| WO2018195129A1 (en) | 2017-04-17 | 2018-10-25 | University Of Maryland, College Park | Embryonic cell cultures and methods of using the same |
| US11834670B2 (en) * | 2017-04-19 | 2023-12-05 | Global Life Sciences Solutions Usa Llc | Site-specific DNA modification using a donor DNA repair template having tandem repeat sequences |
| CN116555353A (zh) | 2017-04-20 | 2023-08-08 | E开创生物技术股份有限公司 | 产生基因修改的动物的方法 |
| US11458118B2 (en) | 2017-04-21 | 2022-10-04 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods and pharmaceutical compositions for the treatment of diseases associated with reduced CFTR function |
| BR112019021719A2 (pt) | 2017-04-21 | 2020-06-16 | The General Hospital Corporation | Variantes de cpf1 (cas12a) com especificidade para pam alterada |
| AU2018254616B2 (en) | 2017-04-21 | 2022-07-28 | The General Hospital Corporation | Inducible, tunable, and multiplex human gene regulation using crispr-Cpf1 |
| EP3615552A1 (en) | 2017-04-24 | 2020-03-04 | DuPont Nutrition Biosciences ApS | Methods and compositions of anti-crispr proteins for use in plants |
| US11499151B2 (en) | 2017-04-28 | 2022-11-15 | Editas Medicine, Inc. | Methods and systems for analyzing guide RNA molecules |
| WO2018201051A1 (en) | 2017-04-28 | 2018-11-01 | Novartis Ag | Bcma-targeting agent, and combination therapy with a gamma secretase inhibitor |
| US20200055948A1 (en) | 2017-04-28 | 2020-02-20 | Novartis Ag | Cells expressing a bcma-targeting chimeric antigen receptor, and combination therapy with a gamma secretase inhibitor |
| US12157883B2 (en) * | 2017-05-05 | 2024-12-03 | California Institute Of Technology | DNA sequence modification-based gene drive |
| CN110997705A (zh) | 2017-05-05 | 2020-04-10 | 巴塞罗那自治大学 | 纳米结构蛋白及其用途 |
| EP3622070A2 (en) | 2017-05-10 | 2020-03-18 | Editas Medicine, Inc. | Crispr/rna-guided nuclease systems and methods |
| EP3621981A2 (en) | 2017-05-12 | 2020-03-18 | CRISPR Therapeutics AG | Materials and methods for engineering cells and uses thereof in immuno-oncology |
| US11560566B2 (en) | 2017-05-12 | 2023-01-24 | President And Fellows Of Harvard College | Aptazyme-embedded guide RNAs for use with CRISPR-Cas9 in genome editing and transcriptional activation |
| WO2018213351A1 (en) * | 2017-05-16 | 2018-11-22 | The Regents Of The University Of California | Thermostable rna-guided endonucleases and methods of use thereof |
| WO2018211018A1 (en) | 2017-05-17 | 2018-11-22 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Flt3 inhibitors for improving pain treatments by opioids |
| US11439692B2 (en) | 2017-05-17 | 2022-09-13 | Modalis Therapeutics Corporation | Method of treating diseases associated with MYD88 pathways using CRISPR-GNDM system |
| WO2018218038A1 (en) | 2017-05-24 | 2018-11-29 | Effector Therapeutics, Inc. | Methods and compositions for cellular immunotherapy |
| US11326157B2 (en) | 2017-05-25 | 2022-05-10 | The General Hospital Corporation | Base editors with improved precision and specificity |
| US11788087B2 (en) | 2017-05-25 | 2023-10-17 | The Children's Medical Center Corporation | BCL11A guide delivery |
| CN108977442B (zh) * | 2017-06-05 | 2023-01-06 | 广州市锐博生物科技有限公司 | 用于dna编辑的系统及其应用 |
| WO2018226560A1 (en) | 2017-06-05 | 2018-12-13 | Regeneron Pharmaceuticals, Inc. | B4galt1 variants and uses thereof |
| CN110719956A (zh) | 2017-06-06 | 2020-01-21 | 齐默尔根公司 | 用于改良真菌菌株的高通量基因组工程改造平台 |
| WO2018226880A1 (en) | 2017-06-06 | 2018-12-13 | Zymergen Inc. | A htp genomic engineering platform for improving escherichia coli |
| WO2018224615A1 (en) | 2017-06-08 | 2018-12-13 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods and compositions for treating hyperpigmentation disorders |
| US11517901B2 (en) | 2017-06-09 | 2022-12-06 | The Regents Of The University Of California | High-efficiency particle encapsulation in droplets with particle spacing and downstream droplet sorting |
| WO2018227210A1 (en) | 2017-06-09 | 2018-12-13 | The Regents Of The University Of California | High-efficiency encapsulation in droplets based on hydrodynamic vortices control |
| JP2020524993A (ja) | 2017-06-13 | 2020-08-27 | フラッグシップ パイオニアリング イノベーションズ ブイ, インコーポレイテッド | クロンを含む組成物及びその使用 |
| US20200362355A1 (en) | 2017-06-15 | 2020-11-19 | The Regents Of The University Of California | Targeted non-viral dna insertions |
| US10011849B1 (en) | 2017-06-23 | 2018-07-03 | Inscripta, Inc. | Nucleic acid-guided nucleases |
| US9982279B1 (en) | 2017-06-23 | 2018-05-29 | Inscripta, Inc. | Nucleic acid-guided nucleases |
| AU2018289077B2 (en) | 2017-06-23 | 2022-03-10 | Inscripta, Inc. | Nucleic acid-guided nucleases |
| CN111051510A (zh) | 2017-06-25 | 2020-04-21 | 斯尼普技术有限公司 | 改变微生物群体和修饰微生物群 |
| CN118109477A (zh) | 2017-06-27 | 2024-05-31 | 瑞泽恩制药公司 | 包含人源化asgr1基因座的非人动物 |
| EP3645721A1 (en) | 2017-06-30 | 2020-05-06 | Novartis AG | Methods for the treatment of disease with gene editing systems |
| US11168322B2 (en) | 2017-06-30 | 2021-11-09 | Arbor Biotechnologies, Inc. | CRISPR RNA targeting enzymes and systems and uses thereof |
| EP4270012A3 (en) | 2017-06-30 | 2024-04-03 | Inscripta, Inc. | Automated cell processing methods, modules, instruments and systems |
| JP2020530307A (ja) | 2017-06-30 | 2020-10-22 | インティマ・バイオサイエンス,インコーポレーテッド | 遺伝子治療のためのアデノ随伴ウイルスベクター |
| JP7588391B2 (ja) | 2017-06-30 | 2024-11-22 | コデクシス, インコーポレイテッド | T7 rnaポリメラーゼバリアント |
| EP3645712A4 (en) | 2017-06-30 | 2021-07-07 | Codexis, Inc. | T7 RNA POLYMERASE VARIANTS |
| TWI821192B (zh) | 2017-07-11 | 2023-11-11 | 美商新索思股份有限公司 | 非天然核苷酸之導入及其方法 |
| EP3652312A1 (en) | 2017-07-14 | 2020-05-20 | Editas Medicine, Inc. | Systems and methods for targeted integration and genome editing and detection thereof using integrated priming sites |
| EP3427756A1 (en) | 2017-07-14 | 2019-01-16 | Universidad Autónoma De Barcelona (UAB) | Therapeutic nanoconjugates and uses thereof |
| US11452967B2 (en) | 2017-07-17 | 2022-09-27 | Zymergen Inc. | Metal-organic framework materials |
| WO2019018551A2 (en) | 2017-07-18 | 2019-01-24 | Lee Tzumin | METHODS AND COMPOSITIONS FOR GENETICALLY GENETIC HANDLING OF GENES AND CELLS |
| WO2019016310A1 (en) | 2017-07-20 | 2019-01-24 | INSERM (Institut National de la Santé et de la Recherche Médicale) | METHODS AND COMPOSITIONS FOR TREATING CANCERS |
| CN109295054B (zh) * | 2017-07-25 | 2024-02-06 | 广州普世利华科技有限公司 | 用于靶向病原体基因RNA的gRNA及基于C2c2的病原体基因的检测方法及试剂盒 |
| US11926664B2 (en) | 2017-07-25 | 2024-03-12 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods and pharmaceutical compositions for modulating monocytopoiesis |
| WO2019023680A1 (en) | 2017-07-28 | 2019-01-31 | President And Fellows Of Harvard College | METHODS AND COMPOSITIONS FOR EVOLUTION OF BASIC EDITORS USING PHAGE-ASSISTED CONTINUOUS EVOLUTION (PACE) |
| KR102712142B1 (ko) | 2017-07-31 | 2024-10-07 | 리제너론 파마슈티칼스 인코포레이티드 | Cas-형질전환 마우스 배아 줄기 세포 및 마우스 및 이것의 용도 |
| JP2020533957A (ja) | 2017-07-31 | 2020-11-26 | リジェネロン・ファーマシューティカルズ・インコーポレイテッドRegeneron Pharmaceuticals, Inc. | Crisprリポーター非ヒト動物およびその使用 |
| KR20200032117A (ko) | 2017-07-31 | 2020-03-25 | 리제너론 파마슈티칼스 인코포레이티드 | 생체 내에서 외인성 공여체 핵산과의 CRISPR/Cas-유도된 재조합의 평가 |
| EP4445954A3 (en) | 2017-07-31 | 2025-01-08 | Reflection Biotechnologies Limited | Cellular models of and therapies for ocular diseases |
| CN111278848B (zh) | 2017-08-04 | 2023-06-27 | 北京大学 | 特异性识别甲基化修饰dna碱基的tale rvd及其应用 |
| US11384383B2 (en) | 2017-08-08 | 2022-07-12 | Depixus | In vitro isolation and enrichment of nucleic acids using site-specific nucleases |
| US11624077B2 (en) | 2017-08-08 | 2023-04-11 | Peking University | Gene knockout method |
| AU2018315731B2 (en) | 2017-08-09 | 2024-10-03 | Ricetec, Inc. | Compositions and methods for modifying genomes |
| WO2019036599A1 (en) | 2017-08-18 | 2019-02-21 | The Board Of Regents Of The University Of Texas System | EXON DELETION CORRECTION OF MUTATIONS OF DUCHENNE MUSCLE DYSTROPHY IN ACTINE DYSTROPHINE BINDING DOMAIN 1 Using a GENOME CRISPR EDITION |
| RU2020111575A (ru) * | 2017-08-22 | 2021-09-23 | Напиджен, Инк. | Модификация генома органелл с использованием направляемой полинуклеотидом эндонуклеазы |
| US11286468B2 (en) | 2017-08-23 | 2022-03-29 | The General Hospital Corporation | Engineered CRISPR-Cas9 nucleases with altered PAM specificity |
| US10738327B2 (en) | 2017-08-28 | 2020-08-11 | Inscripta, Inc. | Electroporation cuvettes for automation |
| US11319532B2 (en) | 2017-08-30 | 2022-05-03 | President And Fellows Of Harvard College | High efficiency base editors comprising Gam |
| WO2019050948A1 (en) * | 2017-09-05 | 2019-03-14 | Regeneron Pharmaceuticals, Inc. | ADMINISTRATION OF A GENE EDITION SYSTEM HAVING ONLY ONE RETROVIRAL PARTICLE AND METHODS OF GENERATING AND USING |
| SG11202001754RA (en) | 2017-09-06 | 2020-03-30 | Regeneron Pharma | Single immunoglobulin interleukin-1 receptor related (sigirr) variants and uses thereof |
| CN111065410A (zh) | 2017-09-06 | 2020-04-24 | 弗雷德哈钦森癌症研究中心 | 用于改善过继细胞疗法的方法 |
| JP7407701B2 (ja) | 2017-09-06 | 2024-01-04 | フレッド ハッチンソン キャンサー センター | strepタグ特異的キメラ受容体およびその使用 |
| EP3679060A1 (en) | 2017-09-07 | 2020-07-15 | Regeneron Pharmaceuticals, Inc. | Solute carrier family 14 member 1 (slc14a1) variants and uses thereof |
| WO2019051097A1 (en) | 2017-09-08 | 2019-03-14 | The Regents Of The University Of California | RNA-GUIDED ENDONUCLEASE FUSION POLYPEPTIDES AND METHODS OF USING SAME |
| US20190076814A1 (en) * | 2017-09-11 | 2019-03-14 | Synthego Corporation | Biopolymer synthesis system and method |
| AU2018329745A1 (en) * | 2017-09-11 | 2020-04-16 | The Regents Of The University Of California | Antibody-mediated delivery of Cas9 to mammalian cells |
| CN110168093B (zh) * | 2017-09-12 | 2023-08-15 | 中科蓝华(广州)生物医药技术有限公司 | 一种转染细胞内寄生虫的试剂盒及其应用 |
| WO2019055862A1 (en) | 2017-09-14 | 2019-03-21 | Fred Hutchinson Cancer Research Center | HIGH AFFINITY T CELL RECEPTORS AND USES THEREOF |
| CA3072614A1 (en) | 2017-09-18 | 2019-03-21 | Futuragene Israel Ltd. | Tissue-specific expression control of della polypeptides |
| WO2019052577A1 (zh) | 2017-09-18 | 2019-03-21 | 博雅辑因(北京)生物科技有限公司 | 一种基因编辑t细胞及其用途 |
| WO2019060469A2 (en) * | 2017-09-19 | 2019-03-28 | Massachusetts Institute Of Technology | CAS9 STREPTOCOCCUS CANIS AS A GENOMIC ENGINEERING PLATFORM WITH NEW PAM SPECIFICITY |
| US11453865B2 (en) * | 2017-09-19 | 2022-09-27 | Massachusetts Institute Of Technology | Applications of engineered Streptococcus canis Cas9 variants on single-base PAM targets |
| KR102794940B1 (ko) | 2017-09-19 | 2025-04-14 | 어드박시스, 인크. | 박테리아 또는 리스테리아 균주의 동결건조를 위한 조성물 및 방법 |
| WO2019057649A1 (en) | 2017-09-19 | 2019-03-28 | INSERM (Institut National de la Santé et de la Recherche Médicale) | METHODS AND PHARMACEUTICAL COMPOSITIONS FOR THE TREATMENT OF ACUTE MYELOID LEUKEMIA |
| KR102742220B1 (ko) | 2017-09-19 | 2024-12-17 | 더 유니버시티 오브 브리티쉬 콜롬비아 | 항-hla-a2 항체 및 그 사용 방법 |
| IT201700105372A1 (it) * | 2017-09-20 | 2019-03-20 | Fondazione St Italiano Tecnologia | Molecola di acido nucleico funzionale e relativo uso |
| WO2019057742A1 (en) | 2017-09-20 | 2019-03-28 | INSERM (Institut National de la Santé et de la Recherche Médicale) | METHODS AND PHARMACEUTICAL COMPOSITIONS FOR MODULATION OF AUTOPHAGIA |
| MX2020003047A (es) | 2017-09-20 | 2020-10-22 | Univ British Columbia | Nuevos anticuerpos anti-hla-a2 y usos de los mismos. |
| BR112020005803A2 (pt) | 2017-09-25 | 2020-12-01 | Agrospheres, Inc. | composições e métodos para produção e administração moduláveis de produtos biológicos |
| US11572574B2 (en) * | 2017-09-28 | 2023-02-07 | Toolgen Incorporated | Artificial genome manipulation for gene expression regulation |
| CN111405912B (zh) | 2017-09-29 | 2025-02-14 | 因特利亚治疗公司 | 用于基因组编辑的多核苷酸、组合物及方法 |
| AU2018338790B2 (en) | 2017-09-29 | 2022-09-15 | Regeneron Pharmaceuticals, Inc. | Non-human animals comprising a humanized TTR locus and methods of use |
| US11845951B2 (en) * | 2017-09-29 | 2023-12-19 | Toolgen Incorporated | Gene manipulation for treatment of retinal dysfunction disorder |
| CN111372650A (zh) | 2017-09-30 | 2020-07-03 | 因思科瑞普特公司 | 流通式电穿孔仪器 |
| CN111447934A (zh) | 2017-10-06 | 2020-07-24 | 4阵营疗法公司 | 用于治疗尿素循环障碍的方法和组合物,尤其是otc缺乏症 |
| JP2020536529A (ja) | 2017-10-10 | 2020-12-17 | チルドレンズ ホスピタル メディカル センター | 食道組織および/または臓器組成物およびそれを作製する方法 |
| KR102694809B1 (ko) | 2017-10-11 | 2024-08-16 | 리제너론 파마슈티칼스 인코포레이티드 | Pnpla3 i148m 변이를 발현하는 환자의 간 질환의 치료에서의 hsd17b13의 저해 |
| US11725228B2 (en) | 2017-10-11 | 2023-08-15 | The General Hospital Corporation | Methods for detecting site-specific and spurious genomic deamination induced by base editing technologies |
| WO2019075409A1 (en) | 2017-10-12 | 2019-04-18 | The Regents Of The University Of California | ISOLATION AND IDENTIFICATION WITHOUT MICROFLUIDIC LABEL OF CELLS USING FLUORESCENCE LIFE IMAGING (FLIM) |
| AU2018352592B2 (en) | 2017-10-16 | 2025-03-27 | Beam Therapeutics, Inc. | Uses of adenosine base editors |
| RU2020115869A (ru) | 2017-10-16 | 2021-11-19 | Ридженерон Фармасьютикалз, Инк. | Варианты корнулина (crnn) и их применение |
| AU2018353012B2 (en) | 2017-10-17 | 2025-03-27 | Bayer Healthcare Llc | Compositions and methods for gene editing for Hemophilia A |
| US11745179B2 (en) * | 2017-10-20 | 2023-09-05 | The Regents Of The University Of California | Microfluidic systems and methods for lipoplex-mediated cell transfection |
| WO2019079777A1 (en) | 2017-10-20 | 2019-04-25 | Fred Hutchinson Cancer Research Center | COMPOSITIONS AND METHODS FOR TIGIT AND / OR CD112R TARGETING IMMUNOTHERAPY OR COMPRISING THE OVEREXPRESSION OF CD226 |
| US11499127B2 (en) | 2017-10-20 | 2022-11-15 | The Regents Of The University Of California | Multi-layered microfluidic systems for in vitro large-scale perfused capillary networks |
| WO2019081428A1 (en) | 2017-10-23 | 2019-05-02 | INSERM (Institut National de la Santé et de la Recherche Médicale) | COMPOUNDS FOR THE TREATMENT OF CMV RELATED DISEASES |
| US12227742B2 (en) | 2017-10-23 | 2025-02-18 | The Broad Institute, Inc. | Nucleic acid modifiers |
| US20210180091A1 (en) | 2017-10-26 | 2021-06-17 | Vertex Pharmaceuticals Incorporated | Materials and methods for treatment of hemoglobinopathies |
| CN109722415B (zh) | 2017-10-27 | 2021-01-26 | 博雅辑因(北京)生物科技有限公司 | 一种造血干细胞的培养组合物、培养基以及造血干细胞的培养方法 |
| CA3080415A1 (en) | 2017-10-27 | 2019-05-02 | The Regents Of The University Of California | Targeted replacement of endogenous t cell receptors |
| US20210179709A1 (en) | 2017-10-31 | 2021-06-17 | Novartis Ag | Anti-car compositions and methods |
| JP2021503278A (ja) | 2017-11-01 | 2021-02-12 | ザ リージェンツ オブ ザ ユニバーシティ オブ カリフォルニア | CasZ組成物及び使用方法 |
| US12227753B2 (en) | 2017-11-01 | 2025-02-18 | The Regents Of The University Of California | CasY compositions and methods of use |
| EP3704245A1 (en) | 2017-11-01 | 2020-09-09 | Novartis AG | Synthetic rnas and methods of use |
| US11970719B2 (en) | 2017-11-01 | 2024-04-30 | The Regents Of The University Of California | Class 2 CRISPR/Cas compositions and methods of use |
| WO2019090173A1 (en) * | 2017-11-02 | 2019-05-09 | Arbor Biotechnologies, Inc. | Novel crispr-associated transposon systems and components |
| WO2019092505A1 (en) | 2017-11-09 | 2019-05-16 | Casebia Therapeutics Llp | Self-inactivating (sin) crispr/cas or crispr/cpf1 systems and uses thereof |
| WO2019092042A1 (en) | 2017-11-10 | 2019-05-16 | Novozymes A/S | Temperature-sensitive cas9 protein |
| WO2019094928A1 (en) | 2017-11-10 | 2019-05-16 | Massachusetts Institute Of Technology | Microbial production of pure single stranded nucleic acids |
| US20190141966A1 (en) | 2017-11-10 | 2019-05-16 | Regeneron Pharmaceuticals, Inc. | Non-Human Animals Comprising SLC30A8 Mutation And Methods Of Use |
| CN111448313A (zh) | 2017-11-16 | 2020-07-24 | 阿斯利康(瑞典)有限公司 | 用于改善基于Cas9的敲入策略的有效性的组合物和方法 |
| EP3713644B1 (en) | 2017-11-20 | 2024-08-07 | University of Georgia Research Foundation, Inc. | Compositions and methods for modulating hif-2a to improve muscle generation and repair |
| WO2019101995A1 (en) | 2017-11-27 | 2019-05-31 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods and pharmaceutical compositions for cardiac regeneration |
| US20190160120A1 (en) * | 2017-11-29 | 2019-05-30 | Snipr Biome Aps | Dna, methods etc |
| CN116064611A (zh) | 2017-11-30 | 2023-05-05 | 瑞泽恩制药公司 | 包含人源化trkb基因座的非人动物 |
| US20220267403A1 (en) | 2017-12-01 | 2022-08-25 | Fred Hutchinson Cancer Research Center | Binding proteins specific for 5t4 and uses thereof |
| WO2019113096A1 (en) * | 2017-12-05 | 2019-06-13 | BioPlx, Inc. | Methods and compositions to prevent microbial infection |
| US12161674B2 (en) | 2017-12-05 | 2024-12-10 | Vertex Pharmaceuticals Incorporated | CRISPR-CAS9 modified CD34+ human hematopoietic stem and progenitor cells and uses thereof |
| EP3720453B1 (en) | 2017-12-05 | 2024-07-10 | Caribou Biosciences, Inc. | Modified lymphocytes |
| EP3720944A1 (en) | 2017-12-07 | 2020-10-14 | Zymergen Inc. | Engineered biosynthetic pathways for production of (6e)-8-hydroxygeraniol by fermentation |
| MX2020005939A (es) | 2017-12-08 | 2020-08-24 | Synthetic Genomics Inc | Mejoramiento de la productividad de lipidos de algas a traves de la modificacion genetica de una proteina que contiene un dominio tpr. |
| US11661599B1 (en) | 2017-12-14 | 2023-05-30 | National Technology & Engineering Solutions Of Sandia, Llc | CRISPR-Cas based system for targeting single-stranded sequences |
| US11766455B2 (en) | 2017-12-14 | 2023-09-26 | Ezy Biotech Llc | Subject-specific tumor inhibiting cells and the use thereof |
| JP2021506251A (ja) * | 2017-12-14 | 2021-02-22 | クリスパー セラピューティクス アーゲー | 新規rnaプログラム可能エンドヌクレアーゼ系、ならびにゲノム編集および他の適用におけるその使用 |
| CA3084824A1 (en) | 2017-12-15 | 2019-06-20 | Flagship Pioneering Innovations Vi, Llc | Compositions comprising circular polyribonucleotides and uses thereof |
| JP7384801B2 (ja) | 2017-12-15 | 2023-11-21 | ダニスコ・ユーエス・インク | Cas9変異体及び使用方法 |
| JP7402163B2 (ja) | 2017-12-21 | 2023-12-20 | クリスパー セラピューティクス アーゲー | 2a型アッシャー症候群の処置のための材料および方法 |
| AU2018395264B2 (en) | 2017-12-29 | 2023-06-22 | Synthetic Genomics, Inc. | Genetic modulation of photosynthetic organisms for improved growth |
| EP3732187A4 (en) | 2017-12-29 | 2021-11-10 | The Scripps Research Institute | PAIRING COMPOSITIONS OF NON-NATURAL BASES AND METHODS OF USE |
| US20190201548A1 (en) | 2017-12-29 | 2019-07-04 | Rubius Therapeutics, Inc. | Gene editing and targeted transcriptional modulation for engineering erythroid cells |
| US12337036B2 (en) | 2018-01-01 | 2025-06-24 | Aposense Ltd | Compounds and methods for trans-membrane delivery of molecules |
| US20210072244A1 (en) | 2018-01-04 | 2021-03-11 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods and compositions for treating melanoma resistant |
| EP3735462A1 (en) | 2018-01-05 | 2020-11-11 | The Board of Regents of The University of Texas System | Therapeutic crispr/cas9 compositions and methods of use |
| CA3087266A1 (en) | 2018-01-09 | 2019-07-18 | Cibus Us Llc | Shatterproof genes and mutations |
| WO2019140278A1 (en) | 2018-01-11 | 2019-07-18 | Fred Hutchinson Cancer Research Center | Immunotherapy targeting core binding factor antigens |
| US20200362366A1 (en) | 2018-01-12 | 2020-11-19 | Basf Se | Gene underlying the number of spikelets per spike qtl in wheat on chromosome 7a |
| US11268092B2 (en) | 2018-01-12 | 2022-03-08 | GenEdit, Inc. | Structure-engineered guide RNA |
| CA3088180A1 (en) | 2018-01-12 | 2019-07-18 | Crispr Therapeutics Ag | Compositions and methods for gene editing by targeting transferrin |
| AU2019209290B2 (en) * | 2018-01-17 | 2024-03-07 | Vertex Pharmaceuticals Incorporated | DNA-PK inhibitors |
| US12269804B2 (en) | 2018-01-17 | 2025-04-08 | Vertex Pharmaceuticals Incorporated | Quinoxalinone compounds, compositions, methods, and kits for increasing genome editing efficiency |
| JP2021510594A (ja) | 2018-01-25 | 2021-04-30 | アンスティチュ ナショナル ドゥ ラ サンテ エ ドゥ ラ ルシェルシュ メディカル | 臓器の虚血再灌流傷害を予防する方法における使用のためのil−33のアンタゴニスト |
| US12098363B2 (en) * | 2018-01-26 | 2024-09-24 | The Children's Medical Center Corporation | Targeting BCL11A distal regulatory elements with a CAS9-CAS9 fusion for fetal hemoglobin reinduction |
| WO2019147275A1 (en) * | 2018-01-26 | 2019-08-01 | Integrated Dna Technologies, Inc. | Crispr-based compositions and methods of use |
| US11926835B1 (en) | 2018-01-29 | 2024-03-12 | Inari Agriculture Technology, Inc. | Methods for efficient tomato genome editing |
| AU2019216257A1 (en) | 2018-01-31 | 2020-07-23 | Research Institute At Nationwide Children's Hospital | Gene therapy for limb-girdle muscular dystrophy type 2C |
| BR112020015617A2 (pt) | 2018-01-31 | 2021-01-05 | The Board Of Regents Of The University Of Texas System | Composições e métodos para correção de mutações da distrofina em cardiomiócitos humanos |
| WO2019150309A1 (en) | 2018-02-02 | 2019-08-08 | Hammack Scott | Modulators of gpr68 and uses thereof for treating and preventing diseases |
| MA51787A (fr) | 2018-02-05 | 2020-12-16 | Vertex Pharma | Substances et méthodes de traitement d'hémoglobinopathies |
| US20210093679A1 (en) | 2018-02-05 | 2021-04-01 | Novome Biotechnologies, Inc. | Engineered gut microbes and uses thereof |
| MA51788A (fr) | 2018-02-05 | 2020-12-16 | Vertex Pharma | Substances et méthodes pour traiter des hémoglobinopathies |
| CN111699254A (zh) | 2018-02-08 | 2020-09-22 | 齐默尔根公司 | 使用crispr在棒状杆菌属中进行基因组编辑 |
| IL274528B1 (en) | 2018-02-15 | 2025-06-01 | Sigma Aldrich Co Llc | Engineered CAS9 systems for eukaryotic genome modification |
| EP3752616A1 (en) | 2018-02-16 | 2020-12-23 | CRISPR Therapeutics AG | Compositions and methods for gene editing by targeting fibrinogen-alpha |
| CA3091311A1 (en) | 2018-02-16 | 2019-08-22 | Inserm (Institut National De La Sante Et De La Recherche Medicale) | Use of antagonists of cxcr3b for treating vitiligo |
| US11718849B2 (en) | 2018-02-19 | 2023-08-08 | Agilent Technologies, Inc. | Phosphopeptide-encoding oligonucleotide libraries and methods for detecting phosphorylation-dependent molecular interactions |
| CA3091267A1 (en) | 2018-02-23 | 2019-08-29 | Pioneer Hi-Bred International, Inc. | Novel cas9 orthologs |
| CN112041335A (zh) | 2018-02-26 | 2020-12-04 | 弗雷德哈钦森癌症研究中心 | 用于细胞免疫治疗的组合物和方法 |
| US11713472B2 (en) | 2018-03-06 | 2023-08-01 | The Board Of Trustees Of The University Of Illinois | Genome editing in Archaea |
| WO2019173684A1 (en) | 2018-03-09 | 2019-09-12 | Advaxis, Inc. | Compositions and methods for evaluating attenuation and infectivity of listeria strains |
| BR112020018646A2 (pt) * | 2018-03-12 | 2020-12-29 | Pioneer Hi-Bred International, Inc. | Métodos para transformação de planta |
| SG11202008956XA (en) | 2018-03-14 | 2020-10-29 | Editas Medicine Inc | Systems and methods for the treatment of hemoglobinopathies |
| JP7558563B2 (ja) | 2018-03-15 | 2024-10-01 | ケーエスキュー セラピューティクス, インコーポレイテッド | 免疫療法の改善のための遺伝子調節組成物及び遺伝子調節方法 |
| JP7550648B2 (ja) * | 2018-03-19 | 2024-09-13 | クリスパー セラピューティクス アーゲー | 新規rnaプログラム可能エンドヌクレアーゼ系およびその使用 |
| CN116349651A (zh) | 2018-03-19 | 2023-06-30 | 瑞泽恩制药公司 | 使用crispr/cas系统对动物进行转录调制 |
| CN111936628B (zh) | 2018-03-20 | 2024-10-18 | 清华大学 | 阿尔茨海默病动物模型及其应用 |
| AU2019239971A1 (en) | 2018-03-21 | 2020-09-03 | Regeneron Pharmaceuticals, Inc. | 17beta-hydroxysteroid dehydrogenase type 13 (HSD17b13) iRNA compositions and methods of use thereof |
| US10760075B2 (en) | 2018-04-30 | 2020-09-01 | Snipr Biome Aps | Treating and preventing microbial infections |
| EP3781196B1 (en) * | 2018-03-27 | 2024-03-06 | G+Flas Life Sciences | Sequence-specific in vivo cell targeting |
| EP3775159A4 (en) | 2018-03-29 | 2022-01-19 | Inscripta, Inc. | AUTOMATED REGULATION OF CELL GROWTH RATES FOR INDUCTION AND TRANSFORMATION |
| WO2019193375A1 (en) | 2018-04-04 | 2019-10-10 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Use of fzd7 inhibitors for the treatment of retinal neovascularization |
| AU2019247403B2 (en) | 2018-04-04 | 2025-04-10 | Cibus Europe, B.V. | FAD2 genes and mutations |
| JP7522038B2 (ja) | 2018-04-06 | 2024-07-24 | ザ チルドレンズ メディカル センター コーポレーション | 体細胞リプログラミングおよびインプリンティングのモジュレートのための組成物および方法 |
| JP7447014B2 (ja) | 2018-04-13 | 2024-03-11 | サンガモ セラピューティクス フランス | インターロイキン23受容体に特異的なキメラ抗原受容体 |
| WO2019200004A1 (en) | 2018-04-13 | 2019-10-17 | Inscripta, Inc. | Automated cell processing instruments comprising reagent cartridges |
| EP3781585A4 (en) | 2018-04-17 | 2022-01-26 | The General Hospital Corporation | SENSITIVE IN VITRO ASSAYS FOR SUBSTRATE AND SITE PREFERENCES OF NUCLEIC ACID BINDERS, MODIFIERS AND CLEAVATORS |
| WO2019204668A1 (en) | 2018-04-18 | 2019-10-24 | Casebia Therapeutics Limited Liability Partnership | Compositions and methods for knockdown of apo(a) by gene editing for treatment of cardiovascular disease |
| SG11202009783WA (en) | 2018-04-19 | 2020-11-27 | Univ California | Compositions and methods for gene editing |
| US10501738B2 (en) | 2018-04-24 | 2019-12-10 | Inscripta, Inc. | Automated instrumentation for production of peptide libraries |
| US10858761B2 (en) | 2018-04-24 | 2020-12-08 | Inscripta, Inc. | Nucleic acid-guided editing of exogenous polynucleotides in heterologous cells |
| US10557216B2 (en) | 2018-04-24 | 2020-02-11 | Inscripta, Inc. | Automated instrumentation for production of T-cell receptor peptide libraries |
| WO2019210057A1 (en) | 2018-04-27 | 2019-10-31 | Seattle Children's Hospital (dba Seattle Children's Research Institute) | Rapamycin resistant cells |
| US20210239681A1 (en) | 2018-04-27 | 2021-08-05 | Advaxis, Inc. | Compositions and methods for evaluating potency of listeria-based immunotherapeutics |
| WO2019210153A1 (en) | 2018-04-27 | 2019-10-31 | Novartis Ag | Car t cell therapies with enhanced efficacy |
| WO2019213013A1 (en) | 2018-05-02 | 2019-11-07 | The Children's Medical Center Corporation | Improved bcl11a micrornas for treating hemoglobinopathies |
| CA3098874A1 (en) | 2018-05-10 | 2019-11-14 | The Board Of Trustees Of The Leland Stanford Junior University | Gene therapy methods and compositions using auxotrophic regulatable cells |
| CN112105732A (zh) * | 2018-05-10 | 2020-12-18 | 先正达参股股份有限公司 | 用于多核苷酸的靶向编辑的方法和组合物 |
| US11904081B2 (en) | 2018-05-11 | 2024-02-20 | Lupagen, Inc. | Systems and methods for closed loop, real-time modifications of patient cells |
| AU2019265019A1 (en) | 2018-05-11 | 2020-11-26 | Beam Therapeutics Inc. | Methods of substituting pathogenic amino acids using programmable base editor systems |
| WO2019222545A1 (en) | 2018-05-16 | 2019-11-21 | Synthego Corporation | Methods and systems for guide rna design and use |
| WO2019222513A1 (en) | 2018-05-17 | 2019-11-21 | Regents Of The University Of Minnesota | Drug-resistant immune cells and methods of use thereof |
| EP3796920A4 (en) | 2018-05-22 | 2023-03-01 | The Regents Of The University Of California | TRNA/PRE-MIRNA COMPOSITIONS AND USE IN THE TREATMENT OF CANCER |
| EP3797160A1 (en) | 2018-05-23 | 2021-03-31 | The Broad Institute Inc. | Base editors and uses thereof |
| US12129485B2 (en) | 2018-05-23 | 2024-10-29 | National University Of Singapore | Blockade of CD2 surface expression and expression of chimeric antigen receptors for immunotherapy of T-cell malignancies |
| EP3572512A1 (en) | 2018-05-24 | 2019-11-27 | B.R.A.I.N. Ag | A method for engineering a protein |
| US11866719B1 (en) | 2018-06-04 | 2024-01-09 | Inari Agriculture Technology, Inc. | Heterologous integration of regulatory elements to alter gene expression in wheat cells and wheat plants |
| CA3102840A1 (en) * | 2018-06-05 | 2019-12-12 | Lifeedit, Inc. | Rna-guided nucleases and active fragments and variants thereof and methods of use |
| WO2019236893A2 (en) * | 2018-06-07 | 2019-12-12 | Allen Institute | Stem cell lines containing endogenous, differentially-expressed tagged proteins, methods of production, and use thereof |
| FR3082208A1 (fr) | 2018-06-11 | 2019-12-13 | Fondation Mediterranee Infection | Methode de modification d'une sequence cible d'acide nucleique d'une cellule hote |
| EP3806962A1 (en) | 2018-06-13 | 2021-04-21 | Novartis AG | Bcma chimeric antigen receptors and uses thereof |
| WO2019246488A1 (en) | 2018-06-21 | 2019-12-26 | Duke University | Compositions and methods for the production of pyruvic acid and related products using dynamic metabolic control |
| AU2019291918B2 (en) | 2018-06-29 | 2025-06-12 | Editas Medicine, Inc. | Synthetic guide molecules, compositions and methods relating thereto |
| EP3814369A1 (en) | 2018-06-29 | 2021-05-05 | Stichting Het Nederlands Kanker Instituut- Antoni van Leeuwenhoek Ziekenhuis | Tweak-receptor agonists for use in combination with immunotherapy of a cancer |
| EP4070802A1 (en) | 2018-06-30 | 2022-10-12 | Inscripta, Inc. | Instruments, modules, and methods for improved detection of edited sequences in live cells |
| WO2020014235A1 (en) | 2018-07-09 | 2020-01-16 | The Regents Of The University Of California | Gene targets for t-cell-based immunotherapy |
| US20210285009A1 (en) | 2018-07-13 | 2021-09-16 | The Regents Of The University Of California | Retrotransposon-based delivery vehicle and methods of use thereof |
| CN113039276A (zh) | 2018-07-16 | 2021-06-25 | 密歇根大学董事会 | 核酸酶介导的核酸修饰 |
| WO2020018964A1 (en) | 2018-07-20 | 2020-01-23 | Fred Hutchinson Cancer Research Center | Compositions and methods for controlled expression of antigen-specific receptors |
| MX2021000965A (es) * | 2018-07-24 | 2021-06-15 | Flagship Pioneering Innovations Vi Llc | Composiciones que comprenden polirribonucleotidos circulares y usos de estas. |
| WO2020028327A1 (en) * | 2018-07-31 | 2020-02-06 | Intellia Therapeutics, Inc. | Compositions and methods for hydroxyacid oxidase 1 ( hao1) gene editing for treating primary hyperoxaluria type 1 (ph1) |
| EP3829652A4 (en) * | 2018-08-01 | 2022-05-11 | University of Maryland, Baltimore | MODULATION OF MTORC1 ACTIVITY AND AUTOPHAGY BY CIB2-RHEB INTERACTION |
| WO2020028617A1 (en) | 2018-08-01 | 2020-02-06 | University Of Georgia Research Foundation, Inc. | Compositions and methods for improving embryo development |
| EP3830301B1 (en) | 2018-08-01 | 2024-05-22 | Mammoth Biosciences, Inc. | Programmable nuclease compositions and methods of use thereof |
| MX2021001525A (es) | 2018-08-07 | 2021-04-19 | Modalis Therapeutics Corp | Activador novedoso de la transcripcion. |
| SG11202101415UA (en) | 2018-08-10 | 2021-03-30 | Sangamo Therapeutics France | New car constructs comprising tnfr2 domains |
| US11142740B2 (en) | 2018-08-14 | 2021-10-12 | Inscripta, Inc. | Detection of nuclease edited sequences in automated modules and instruments |
| US10752874B2 (en) | 2018-08-14 | 2020-08-25 | Inscripta, Inc. | Instruments, modules, and methods for improved detection of edited sequences in live cells |
| US10532324B1 (en) | 2018-08-14 | 2020-01-14 | Inscripta, Inc. | Instruments, modules, and methods for improved detection of edited sequences in live cells |
| JP2021533773A (ja) | 2018-08-15 | 2021-12-09 | ザイマージェン インコーポレイテッド | ハイスループット代謝操作におけるCRISPRiの適用 |
| US12150967B2 (en) | 2018-08-18 | 2024-11-26 | Seed Health, Inc. | Methods and compositions for honey bee health |
| US20210317429A1 (en) * | 2018-08-20 | 2021-10-14 | The Broad Institute, Inc. | Methods and compositions for optochemical control of crispr-cas9 |
| WO2020041501A1 (en) | 2018-08-22 | 2020-02-27 | Fred Hutchinson Cancer Research Center | Immunotherapy targeting kras or her2 antigens |
| EP3841204A4 (en) | 2018-08-23 | 2022-05-18 | Sangamo Therapeutics, Inc. | CHANGED TARGET SPECIFIC BASE EDITORS |
| EP3844266A1 (en) | 2018-08-28 | 2021-07-07 | Fred Hutchinson Cancer Research Center | Methods and compositions for adoptive t cell therapy incorporating induced notch signaling |
| BR112021003670A2 (pt) | 2018-08-28 | 2021-05-18 | Vor Biopharma, Inc. | células-tronco hematopoéticas geneticamente modificadas e seus usos |
| JP7682538B2 (ja) | 2018-08-30 | 2025-05-26 | ザ ユニバーシティ オブ ノース カロライナ アット チャペル ヒル | フィードバック使用可能合成遺伝子、標的シードマッチカセット、およびその使用 |
| EP3844263A4 (en) | 2018-08-30 | 2022-05-25 | Inscripta, Inc. | ENHANCED DETECTION OF NUCLEASE-EDITED SEQUENCES IN AUTOMATED MODULES AND INSTRUMENTS |
| US11479762B1 (en) | 2018-08-31 | 2022-10-25 | Inari Agriculture Technology, Inc. | Compositions, systems, and methods for genome editing |
| WO2020047353A1 (en) | 2018-08-31 | 2020-03-05 | Yale University | Compositions and methods for enhancing triplex and nuclease-based gene editing |
| WO2020048942A1 (en) | 2018-09-04 | 2020-03-12 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods and pharmaceutical compositions for enhancing cytotoxic t lymphocyte-dependent immune responses |
| JP7444858B2 (ja) | 2018-09-05 | 2024-03-06 | アンスティチュ ナショナル ドゥ ラ サンテ エ ドゥ ラ ルシェルシュ メディカル | 喘息及びアレルギー性疾患を処置するための方法及び組成物 |
| US20210198642A1 (en) | 2018-09-07 | 2021-07-01 | Astrazeneca Ab | Compositions and methods for improved nucleases |
| US20200080107A1 (en) | 2018-09-07 | 2020-03-12 | Crispr Therapeutics Ag | Universal donor cells |
| EP3849545A1 (en) | 2018-09-10 | 2021-07-21 | Institut National de la Santé et de la Recherche Médicale (INSERM) | Methods for the treatment of neurofibromatosis |
| JP7222075B2 (ja) | 2018-09-13 | 2023-02-14 | リジェネロン・ファーマシューティカルズ・インコーポレイテッド | C3糸球体症のモデルとしての補体因子h遺伝子ノックアウトラット |
| RU2706298C1 (ru) * | 2018-09-14 | 2019-11-15 | Закрытое Акционерное Общество "Биокад" | НУКЛЕАЗА PaCas9 |
| WO2020061591A1 (en) | 2018-09-21 | 2020-03-26 | President And Fellows Of Harvard College | Methods and compositions for treating diabetes, and methods for enriching mrna coding for secreted proteins |
| WO2020068702A1 (en) | 2018-09-24 | 2020-04-02 | Fred Hutchinson Cancer Research Center | Chimeric receptor proteins and uses thereof |
| WO2020064702A1 (en) | 2018-09-25 | 2020-04-02 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Use of antagonists of th17 cytokines for the treatment of bronchial remodeling in patients suffering from allergic asthma |
| GB201815820D0 (en) | 2018-09-28 | 2018-11-14 | Univ Wageningen | Off-target activity inhibitors for guided endonucleases |
| WO2020070062A1 (en) | 2018-10-01 | 2020-04-09 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Use of tim-3 inhibitors for the treatment of exacerbations in patients suffering from severe asthma |
| EP3861120A4 (en) | 2018-10-01 | 2023-08-16 | North Carolina State University | Recombinant type i crispr-cas system |
| WO2020072254A1 (en) | 2018-10-01 | 2020-04-09 | North Carolina State University | Recombinant type i crispr-cas system and uses thereof for killing target cells |
| US12264313B2 (en) | 2018-10-01 | 2025-04-01 | North Carolina State University | Recombinant type I CRISPR-Cas system and uses thereof for genome modification and alteration of expression |
| US12203123B2 (en) | 2018-10-01 | 2025-01-21 | North Carolina State University | Recombinant type I CRISPR-Cas system and uses thereof for screening for variant cells |
| JP7288915B2 (ja) | 2018-10-04 | 2023-06-08 | 株式会社カネカ | 植物のゲノム編集に用いられるdna構築物 |
| JP2022512648A (ja) | 2018-10-09 | 2022-02-07 | アンスティチュ ナショナル ドゥ ラ サンテ エ ドゥ ラ ルシェルシュ メディカル | 心筋線維化の処置のためのαV-インテグリン(CD51)阻害剤の使用 |
| WO2020076976A1 (en) | 2018-10-10 | 2020-04-16 | Readcoor, Inc. | Three-dimensional spatial molecular indexing |
| US11851663B2 (en) | 2018-10-14 | 2023-12-26 | Snipr Biome Aps | Single-vector type I vectors |
| CN113348247A (zh) | 2018-10-15 | 2021-09-03 | 泰莱托恩基金会 | 基因组编辑方法与构建体 |
| CA3109984A1 (en) | 2018-10-16 | 2020-04-23 | Pioneer Hi-Bred International, Inc. | Genome edited fine mapping and causal gene identification |
| CA3116885A1 (en) | 2018-10-17 | 2020-04-23 | Crispr Therapeutics Ag | Compositions and methods for delivering transgenes |
| TW202028461A (zh) | 2018-10-18 | 2020-08-01 | 美商英特利亞醫療公司 | 核酸構築體及使用方法 |
| WO2020082047A1 (en) | 2018-10-18 | 2020-04-23 | Intellia Therapeutics, Inc. | Compositions and methods for treating alpha-1 antitrypsin deficiencey |
| WO2020079162A1 (en) | 2018-10-18 | 2020-04-23 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods for inducing full ablation of hematopoiesis |
| SG11202103734PA (en) | 2018-10-18 | 2021-05-28 | Intellia Therapeutics Inc | Compositions and methods for expressing factor ix |
| TW202507011A (zh) | 2018-10-18 | 2025-02-16 | 美商英特利亞醫療公司 | 用於從白蛋白基因座表現轉殖基因的組成物及方法 |
| US11214781B2 (en) | 2018-10-22 | 2022-01-04 | Inscripta, Inc. | Engineered enzyme |
| CN113227368B (zh) | 2018-10-22 | 2023-07-07 | 因思科瑞普特公司 | 工程化酶 |
| US11946047B2 (en) * | 2018-10-23 | 2024-04-02 | Texas Tech University System | Treatment strategies against anthrax by interfering with critical host factors |
| UY38427A (es) | 2018-10-26 | 2020-05-29 | Novartis Ag | Métodos y composiciones para terapia con células oculares |
| US11407995B1 (en) | 2018-10-26 | 2022-08-09 | Inari Agriculture Technology, Inc. | RNA-guided nucleases and DNA binding proteins |
| US12281338B2 (en) | 2018-10-29 | 2025-04-22 | The Broad Institute, Inc. | Nucleobase editors comprising GeoCas9 and uses thereof |
| US20210388389A1 (en) | 2018-10-30 | 2021-12-16 | Yale University | Compositions and methods for rapid and modular generation of chimeric antigen receptor t cells |
| WO2020092704A1 (en) | 2018-10-31 | 2020-05-07 | Zymergen Inc. | Multiplexed deterministic assembly of dna libraries |
| US20220010321A1 (en) | 2018-11-01 | 2022-01-13 | Keygene N.V. | Dual guide rna for crispr/cas genome editing in plants cells |
| US11434477B1 (en) | 2018-11-02 | 2022-09-06 | Inari Agriculture Technology, Inc. | RNA-guided nucleases and DNA binding proteins |
| BR112021008973A8 (pt) | 2018-11-09 | 2023-05-02 | Juno Therapeutics Inc | Receptores de células t específicos para mesotelina e uso dos mesmos em imunoterapia |
| US12194327B2 (en) | 2018-11-14 | 2025-01-14 | University Of Florida Research Foundation, Inc. | Materials and methods for modifying Wolbachia and paratransformation of arthropods |
| EP3880255A1 (en) | 2018-11-16 | 2021-09-22 | Astellas Pharma Inc. | Method for treating muscular dystrophy by targeting utrophin gene |
| RU2712497C1 (ru) * | 2018-11-26 | 2020-01-29 | Автономная некоммерческая образовательная организация высшего образования Сколковский институт науки и технологий | Средство разрезания ДНК на основе Cas9 белка из биотехнологически значимой бактерии Clostridium cellulolyticum |
| EP3887538A1 (en) | 2018-11-28 | 2021-10-06 | Keygene N.V. | Targeted enrichment by endonuclease protection |
| CA3121191A1 (en) | 2018-11-28 | 2020-06-04 | Crispr Therapeutics Ag | Optimized mrna encoding cas9 for use in lnps |
| JP2022513159A (ja) * | 2018-11-29 | 2022-02-07 | フラッグシップ パイオニアリング イノベーションズ ブイ, インコーポレイテッド | Rnaを調節する方法 |
| WO2020112195A1 (en) | 2018-11-30 | 2020-06-04 | Yale University | Compositions, technologies and methods of using plerixafor to enhance gene editing |
| KR20200071198A (ko) | 2018-12-10 | 2020-06-19 | 네오이뮨텍, 인코퍼레이티드 | Nrf2 발현 조절 기반 T 세포 항암면역치료법 |
| US20220025405A1 (en) * | 2018-12-12 | 2022-01-27 | Kyushu University, National University Corporation | Production method for genome-edited cells |
| US11166996B2 (en) | 2018-12-12 | 2021-11-09 | Flagship Pioneering Innovations V, Inc. | Anellovirus compositions and methods of use |
| CN113166744B (zh) | 2018-12-14 | 2025-02-07 | 先锋国际良种公司 | 用于基因组编辑的新颖crispr-cas系统 |
| AU2019406778A1 (en) * | 2018-12-17 | 2021-07-22 | Massachusetts Institute Of Technology | Crispr-associated transposase systems and methods of use thereof |
| EP3898947A1 (en) | 2018-12-19 | 2021-10-27 | King's College London | Immunotherapeutic methods and compositions |
| KR20210105914A (ko) | 2018-12-20 | 2021-08-27 | 리제너론 파마슈티칼스 인코포레이티드 | 뉴클레아제-매개 반복부 팽창 |
| AU2019406214A1 (en) | 2018-12-21 | 2021-08-05 | Northwestern University | Use of annexins in preventing and treating muscle membrane injury |
| AU2019417720A1 (en) * | 2018-12-24 | 2021-07-08 | Visterra, Inc. | Methods for identifying epitopes and paratopes |
| WO2020136216A1 (en) | 2018-12-27 | 2020-07-02 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods of identifying subjects having or at risk of having a coagulation related disorder |
| KR20210149686A (ko) | 2018-12-27 | 2021-12-09 | 라이프에디트 테라퓨틱스, 인크. | 유전자 편집에 유용한 폴리펩티드 및 사용 방법 |
| JP2022515639A (ja) * | 2018-12-31 | 2022-02-21 | エイチティージー モレキュラー ダイアグノスティクス, インコーポレイテッド | 同じ試料中のdnaおよびrnaを検出する方法 |
| KR20220008253A (ko) | 2019-01-03 | 2022-01-20 | 엥스띠뛰 나씨오날 드 라 쌍떼 에 드 라 흐쉐르슈 메디깔 (인쎄름) | 암을 앓는 대상에서 cd8+ t 세포 의존성 면역 반응을 향상시키기 위한 방법 및 약학적 조성물 |
| WO2020142754A2 (en) | 2019-01-04 | 2020-07-09 | Mammoth Biosciences, Inc. | Programmable nuclease improvements and compositions and methods for nucleic acid amplification and detection |
| TWI852977B (zh) * | 2019-01-10 | 2024-08-21 | 美商健生生物科技公司 | 前列腺新抗原及其用途 |
| EP3911746A1 (en) | 2019-01-14 | 2021-11-24 | Institut National de la Santé et de la Recherche Médicale (INSERM) | Methods and kits for generating and selecting a variant of a binding protein with increased binding affinity and/or specificity |
| MX2021008494A (es) | 2019-01-15 | 2021-08-19 | Seminis Vegetable Seeds Inc | Plantas de frijol verde con mejorada resistencia a enfermedades. |
| US20220119516A1 (en) | 2019-01-16 | 2022-04-21 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Variants of erythroferrone and their use |
| US20220072084A1 (en) | 2019-01-17 | 2022-03-10 | Universitat Autonoma De Barcelona (Uab) | Therapeutic nanoconjugates and uses thereof |
| WO2020154500A1 (en) | 2019-01-23 | 2020-07-30 | The Broad Institute, Inc. | Supernegatively charged proteins and uses thereof |
| EP3914708A1 (en) | 2019-01-24 | 2021-12-01 | Massachusetts Institute Of Technology | Nucleic acid nanostructure platform for antigen presentation and vaccine formulations formed therefrom |
| EP3918059A4 (en) * | 2019-01-29 | 2022-11-30 | Flagship Pioneering Innovations V, Inc. | COMPOSITIONS CONTAINING AN ENDONUCLEASIS AND METHODS OF PURIFICATION OF AN ENDONUCLEASE |
| WO2020163396A1 (en) | 2019-02-04 | 2020-08-13 | The General Hospital Corporation | Adenine dna base editor variants with reduced off-target rna editing |
| US20220117911A1 (en) | 2019-02-04 | 2022-04-21 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods and compositions for modulating blood-brain barrier |
| US20220145330A1 (en) | 2019-02-10 | 2022-05-12 | The J. David Gladstone Institutes, a testamentary trust established under the Will of J. David Glads | Modified mitochondrion and methods of use thereof |
| SG11202108812SA (en) | 2019-02-15 | 2021-09-29 | Sigma Aldrich Co Llc | Crispr/cas fusion proteins and systems |
| MA54951A (fr) | 2019-02-15 | 2021-12-22 | Bayer Healthcare Llc | Édition de gène pour l'hémophilie a avec une expression de facteur viii améliorée |
| WO2020169472A2 (en) | 2019-02-18 | 2020-08-27 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods of inducing phenotypic changes in macrophages |
| GB201902277D0 (en) | 2019-02-19 | 2019-04-03 | King S College London | Therapeutic agents |
| WO2020172332A1 (en) | 2019-02-20 | 2020-08-27 | Fred Hutchinson Cancer Research Center | Binding proteins specific for ras neoantigens and uses thereof |
| WO2020176389A1 (en) | 2019-02-25 | 2020-09-03 | Caribou Biosciences, Inc. | Plasmids for gene editing |
| US20220049273A1 (en) * | 2019-03-01 | 2022-02-17 | Arbor Biotechnologies, Inc. | Novel crispr dna targeting enzymes and systems |
| EP3935184B1 (en) | 2019-03-04 | 2024-09-11 | King Abdullah University Of Science And Technology | Compositions and methods of targeted nucleic acid enrichment by loop adapter protection and exonuclease digestion |
| DE212020000516U1 (de) | 2019-03-07 | 2022-01-17 | The Regents of the University of California | CRISPR-CAS-Effektorpolypeptide |
| US11053515B2 (en) | 2019-03-08 | 2021-07-06 | Zymergen Inc. | Pooled genome editing in microbes |
| CA3129871A1 (en) | 2019-03-08 | 2020-09-17 | Zymergen Inc. | Iterative genome editing in microbes |
| US20220145333A1 (en) | 2019-03-11 | 2022-05-12 | Sorrento Therapeutics, Inc. | Improved process for integration of dna constructs using rna-guided endonucleases |
| US20220160764A1 (en) | 2019-03-11 | 2022-05-26 | Fred Hutchinson Cancer Research Center | High avidity wt1 t cell receptors and uses thereof |
| AU2020239225A1 (en) * | 2019-03-12 | 2021-09-30 | Bayer Healthcare Llc | Novel high fidelity RNA-programmable endonuclease systems and uses thereof |
| US11781131B2 (en) | 2019-03-18 | 2023-10-10 | Regeneron Pharmaceuticals, Inc. | CRISPR/Cas dropout screening platform to reveal genetic vulnerabilities associated with tau aggregation |
| ES2970541T3 (es) | 2019-03-18 | 2024-05-29 | Regeneron Pharma | Plataforma de cribado CRISPR/CAS para identificar modificadores genéticos de la siembra o agregación de tau |
| JP7669281B2 (ja) | 2019-03-19 | 2025-04-28 | ザ ブロード インスティテュート,インコーポレーテッド | 編集ヌクレオチド配列を編集するための方法および組成物 |
| AU2020247900A1 (en) | 2019-03-25 | 2021-11-04 | Inscripta, Inc. | Simultaneous multiplex genome editing in yeast |
| US11001831B2 (en) | 2019-03-25 | 2021-05-11 | Inscripta, Inc. | Simultaneous multiplex genome editing in yeast |
| CA3128376A1 (en) | 2019-03-27 | 2020-10-01 | Pioneer Hi-Bred International, Inc. | Plant explant transformation |
| WO2020193740A1 (en) | 2019-03-28 | 2020-10-01 | INSERM (Institut National de la Santé et de la Recherche Médicale) | New strategy for treating pancreatic cancer |
| EP3946330A1 (en) | 2019-03-29 | 2022-02-09 | Institut National de la Santé et de la Recherche Médicale (INSERM) | Methods for the treatment of keloid, hypertrophic scars and/or hyperpigmentation disorders |
| WO2020201362A2 (en) | 2019-04-02 | 2020-10-08 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods of predicting and preventing cancer in patients having premalignant lesions |
| KR20210148154A (ko) | 2019-04-03 | 2021-12-07 | 리제너론 파마슈티칼스 인코포레이티드 | 세이프 하버 좌위 내로의 항체 코딩 서열의 삽입을 위한 방법 및 조성물 |
| IL286905B2 (en) | 2019-04-04 | 2024-06-01 | Regeneron Pharma | Non-human animals containing the human coagulation factor 12 locus |
| ES2923629T3 (es) | 2019-04-04 | 2022-09-29 | Regeneron Pharma | Métodos para la introducción sin cicatrices de modificaciones dirigidas en vectores de direccionamiento |
| US20220162621A1 (en) | 2019-04-05 | 2022-05-26 | Danisco Us Inc | Methods For Polynucleotide Integration Into The Genome Of Bacillus Using Dual Circular Recombinant DNA Constructs And Compositions Thereof |
| WO2020204159A1 (ja) | 2019-04-05 | 2020-10-08 | 国立大学法人大阪大学 | ノックイン細胞の作製方法 |
| WO2020206202A1 (en) | 2019-04-05 | 2020-10-08 | Danisco Us Inc | Methods for integrating a donor dna sequence into the genome of bacillus using linear recombinant dna constructs and compositions thereof |
| WO2020208082A1 (en) | 2019-04-09 | 2020-10-15 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Method for treating cmv related diseases |
| EP4530351A3 (en) | 2019-04-12 | 2025-06-18 | Astrazeneca AB | Compositions and methods for improved gene editing |
| GB201905360D0 (en) | 2019-04-16 | 2019-05-29 | Univ Nottingham | Fungal strains, production and uses thereof |
| EP3959320A1 (en) | 2019-04-24 | 2022-03-02 | Novartis AG | Compositions and methods for selective protein degradation |
| JP2022531269A (ja) | 2019-04-30 | 2022-07-06 | 博雅▲輯▼因(北京)生物科技有限公司 | 異常ヘモグロビン症の治療の有効性を予測するための方法 |
| US20220220565A1 (en) | 2019-04-30 | 2022-07-14 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods and compositions for treating melanoma |
| CN114127274A (zh) * | 2019-04-30 | 2022-03-01 | 埃门多生物公司 | 新型omni-50 crispr核酸酶 |
| US11692197B2 (en) | 2019-05-06 | 2023-07-04 | Inari Agriculture Technology, Inc. | Delivery of biological molecules to plant cells |
| EP3966327A1 (en) | 2019-05-08 | 2022-03-16 | Vertex Pharmaceuticals Incorporated | Crispr/cas all-in-two vector systems for treatment of dmd |
| CA3138329A1 (en) | 2019-05-10 | 2020-11-19 | Basf Se | Regulatory nucleic acid molecules for enhancing gene expression in plants |
| CA3141732A1 (en) | 2019-05-23 | 2020-11-26 | Vor Biopharma Inc | Compositions and methods for cd33 modification |
| WO2020239623A1 (en) | 2019-05-24 | 2020-12-03 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Use of ngal inhibitors for the treating chronic wound |
| AU2020284016A1 (en) | 2019-05-29 | 2021-12-23 | Monsanto Technology Llc | Methods and compositions for generating dominant alleles using genome editing |
| WO2020245208A1 (en) | 2019-06-04 | 2020-12-10 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Use of cd9 as a biomarker and as a biotarget in glomerulonephritis or glomerulosclerosis |
| JP2022535413A (ja) | 2019-06-04 | 2022-08-08 | アンスティチュ ナショナル ドゥ ラ サンテ エ ドゥ ラ ルシェルシュ メディカル | がんの処置のためのp38アルファ-キナーゼ阻害剤と組み合わせたニューロピリンアンタゴニスト |
| KR20220016869A (ko) | 2019-06-04 | 2022-02-10 | 리제너론 파마슈티칼스 인코포레이티드 | 베타-슬립 돌연변이를 갖는 인간화 ttr 좌위를 포함하는 비-인간 동물 및 사용 방법 |
| US10837021B1 (en) | 2019-06-06 | 2020-11-17 | Inscripta, Inc. | Curing for recursive nucleic acid-guided cell editing |
| CA3142883A1 (en) | 2019-06-07 | 2020-12-10 | Benjamin OAKES | Engineered casx systems |
| BR112021022722A2 (pt) | 2019-06-07 | 2022-01-04 | Regeneron Pharma | Animal não humano, célula de animal não humana, genoma de animal não humano, gene de albumina animal não humana humanizada, vetor de alvejamento, método de avaliação da atividade de um reagente, e, método de otimização da atividade de um reagente |
| WO2020249769A1 (en) | 2019-06-14 | 2020-12-17 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods and compositions for treating ocular diseases related to mitochondrial dna maintenance |
| JP7664854B2 (ja) | 2019-06-14 | 2025-04-18 | リジェネロン・ファーマシューティカルズ・インコーポレイテッド | タウオパチーのモデル |
| US10907125B2 (en) | 2019-06-20 | 2021-02-02 | Inscripta, Inc. | Flow through electroporation modules and instrumentation |
| US10920189B2 (en) | 2019-06-21 | 2021-02-16 | Inscripta, Inc. | Genome-wide rationally-designed mutations leading to enhanced lysine production in E. coli |
| US10927385B2 (en) | 2019-06-25 | 2021-02-23 | Inscripta, Inc. | Increased nucleic-acid guided cell editing in yeast |
| US11905532B2 (en) | 2019-06-25 | 2024-02-20 | Massachusetts Institute Of Technology | Compositions and methods for molecular memory storage and retrieval |
| CN114630910A (zh) | 2019-06-25 | 2022-06-14 | 伊纳瑞农业技术有限公司 | 改善的同源依赖修复基因组编辑 |
| CN114008193A (zh) | 2019-06-27 | 2022-02-01 | 瑞泽恩制药公司 | Tdp-43蛋白病的建模 |
| EP3993786A1 (en) | 2019-07-02 | 2022-05-11 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods for the prophylactic treatment of cancer in patients suffering from pancreatitis |
| WO2021001431A1 (en) | 2019-07-02 | 2021-01-07 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Use of pi3ka-selective inhibitors for treating metastatic disease in patients suffering from pancreatic cancer |
| GB201909597D0 (en) | 2019-07-03 | 2019-08-14 | Univ Wageningen | Crispr type v-u1 system from mycobacterium mucogenicum and uses thereof |
| CN114144203A (zh) | 2019-07-12 | 2022-03-04 | 国立研究开发法人理化学研究所 | 源于显性变异基因的疾患的治疗剂 |
| US10653731B1 (en) | 2019-07-15 | 2020-05-19 | Vigene Biosciences Inc. | Recombinantly-modified adeno-associated virus (rAAV) having improved packaging efficiency |
| US10557149B1 (en) | 2019-07-15 | 2020-02-11 | Vigene Biosciences, Inc. | Recombinantly-modified adeno-associated virus helper vectors and their use to improve the packaging efficiency of recombinantly-modified adeno-associated virus |
| US10801042B1 (en) | 2019-07-15 | 2020-10-13 | Vigene Biosciences, Inc. | Use of ion concentrations to increase the packaging efficiency of recombinant adeno-associated virus |
| US20220380801A1 (en) | 2019-07-15 | 2022-12-01 | Medimmune Limited | Tripartite systems for protein dimerization and methods of use |
| WO2021009299A1 (en) | 2019-07-17 | 2021-01-21 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Bcl-xl:fkbp12 fusion proteins suitable for screening agents capable of slowing down the aging process |
| WO2021013911A1 (en) | 2019-07-24 | 2021-01-28 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Inhibitors of the sting pathway for the treatment of hidradenitis suppurativa |
| EP4007584A1 (en) | 2019-08-02 | 2022-06-08 | Institut National de la Santé et de la Recherche Médicale (INSERM) | Neutralizing granzyme b for providing cardioprotection in a subject who experienced a myocardial infarction |
| US12179199B2 (en) | 2019-08-09 | 2024-12-31 | The Regents Of The University Of California | Microfluidic single-cell pairing array for studying cell-cell interactions in isolated compartments |
| WO2021028359A1 (en) | 2019-08-09 | 2021-02-18 | Sangamo Therapeutics France | Controlled expression of chimeric antigen receptors in t cells |
| AU2020329912A1 (en) * | 2019-08-12 | 2022-03-24 | LifeEDIT Therapeutics, Inc. | RNA-guided nucleases and active fragments and variants thereof and methods of use |
| KR20220050176A (ko) | 2019-08-20 | 2022-04-22 | 프레드 헛친슨 켄서 리서치 센터 | Wt-1에 특이적인 t-세포 면역요법 |
| AU2020331987A1 (en) | 2019-08-21 | 2022-03-31 | Research Institute At Nationwide Children's Hospital | Adeno-associated virus vector delivery of alpha-sarcoglycan and the treatment of muscular dystrophy |
| EP4022063A1 (en) | 2019-08-28 | 2022-07-06 | Vor Biopharma Inc. | Compositions and methods for cll1 modification |
| US20220288176A1 (en) * | 2019-08-28 | 2022-09-15 | The Board Of Trustees Of The Leland Stanford Junior University | Circular rna modification and methods of use |
| CN114787352A (zh) | 2019-08-28 | 2022-07-22 | Vor生物制药股份有限公司 | 用于cd123修饰的组合物和方法 |
| CN115151275A (zh) | 2019-08-30 | 2022-10-04 | 耶鲁大学 | 用于向细胞递送核酸的组合物和方法 |
| US20220333133A1 (en) | 2019-09-03 | 2022-10-20 | Voyager Therapeutics, Inc. | Vectorized editing of nucleic acids to correct overt mutations |
| CN114269941A (zh) | 2019-09-04 | 2022-04-01 | 博雅辑因(北京)生物科技有限公司 | 基于脱靶评估评价基因编辑治疗的方法 |
| EP4025712A1 (en) | 2019-09-05 | 2022-07-13 | Institut National de la Santé et de la Recherche Médicale (INSERM) | Method of treatment and pronostic of acute myeloid leukemia |
| JP2022547505A (ja) | 2019-09-05 | 2022-11-14 | クリスパー セラピューティクス アクチェンゲゼルシャフト | ユニバーサルドナー細胞 |
| EP4025697A1 (en) | 2019-09-05 | 2022-07-13 | Benson Hill, Inc. | Compositions and methods for modifying genomes |
| MX2022002663A (es) | 2019-09-05 | 2022-04-07 | Crispr Therapeutics Ag | Celulas donantes universales. |
| CN114729343A (zh) * | 2019-09-10 | 2022-07-08 | 科学方案有限责任公司 | 新的2类ii型和v型crispr-cas rna指导的内切核酸酶 |
| US20230203515A1 (en) | 2019-09-12 | 2023-06-29 | Basf Se | Regulatory Nucleic Acid Molecules for Enhancing Gene Expression in Plants |
| WO2021047775A1 (en) | 2019-09-12 | 2021-03-18 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Use of inhibitors of tgfb/activinb signaling pathway for the treatment of patients suffering from medulloblastoma group 3 |
| WO2021050940A1 (en) | 2019-09-13 | 2021-03-18 | Regeneron Pharmaceuticals, Inc. | Transcription modulation in animals using crispr/cas systems delivered by lipid nanoparticles |
| EP4041894A1 (en) | 2019-09-23 | 2022-08-17 | Omega Therapeutics, Inc. | COMPOSITIONS AND METHODS FOR MODULATING HEPATOCYTE NUCLEAR FACTOR 4-ALPHA (HNF4a) GENE EXPRESSION |
| EP4048807A1 (en) | 2019-09-23 | 2022-08-31 | Omega Therapeutics, Inc. | Compositions and methods for modulating apolipoprotein b (apob) gene expression |
| SG10202008262UA (en) | 2019-09-26 | 2021-04-29 | Seminis Vegetable Seeds Inc | Lettuce plants having resistance to nasonovia ribisnigri biotype nr:1 |
| WO2021063968A1 (en) | 2019-09-30 | 2021-04-08 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Method and composition for diagnosing chronic obstructive pulmonary disease |
| US20210095273A1 (en) * | 2019-09-30 | 2021-04-01 | Sigma-Aldrich Co. Llc | Modulation of microbiota compositions using targeted nucleases |
| EP4038190A1 (en) | 2019-10-03 | 2022-08-10 | Artisan Development Labs, Inc. | Crispr systems with engineered dual guide nucleic acids |
| EP4037714A1 (en) | 2019-10-03 | 2022-08-10 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods and compositions for modulating macrophages polarization |
| WO2021069387A1 (en) | 2019-10-07 | 2021-04-15 | Basf Se | Regulatory nucleic acid molecules for enhancing gene expression in plants |
| WO2021070494A1 (ja) | 2019-10-11 | 2021-04-15 | 住友化学株式会社 | 核酸オリゴマーの製造方法 |
| EP3808766A1 (en) | 2019-10-15 | 2021-04-21 | Sangamo Therapeutics France | Chimeric antigen receptor specific for interleukin-23 receptor |
| US20230174972A1 (en) * | 2019-10-15 | 2023-06-08 | Agency For Science, Technology And Research | Assays For Measuring Nucleic Acid Modifying Enzyme Activity |
| WO2021078359A1 (en) | 2019-10-21 | 2021-04-29 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Use of inhibitors of cubilin for the treatment of chronic kidney diseases |
| WO2021081264A1 (en) | 2019-10-24 | 2021-04-29 | Pairwise Plants Services, Inc. | Optimized crispr-cas nucleases and base editors and methods of use thereof |
| US20240122938A1 (en) | 2019-10-29 | 2024-04-18 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods and compositions for treating uveal melanoma |
| WO2021092513A1 (en) | 2019-11-08 | 2021-05-14 | Regeneron Pharmaceuticals, Inc. | Crispr and aav strategies for x-linked juvenile retinoschisis therapy |
| MX2022005818A (es) * | 2019-11-13 | 2022-08-10 | Crispr Therapeutics Ag | Proceso de fabricacion para producir linfocitos t que expresan receptores de antigenos quimericos. |
| EP4058559A1 (en) * | 2019-11-13 | 2022-09-21 | CRISPR Therapeutics AG | Methods of manufacturing car-t cells |
| WO2021102059A1 (en) | 2019-11-19 | 2021-05-27 | Inscripta, Inc. | Methods for increasing observed editing in bacteria |
| WO2021101950A1 (en) | 2019-11-19 | 2021-05-27 | Danisco Us Inc | Selection marker free methods for modifying the genome of bacillus and compositions thereof |
| WO2021102139A1 (en) | 2019-11-20 | 2021-05-27 | Corbion Biotech, Inc. | Sucrose invertase variants |
| CN110938659B (zh) * | 2019-11-22 | 2022-05-10 | 广东省微生物研究所(广东省微生物分析检测中心) | 一种提高纤维堆囊菌埃博霉素产量的dCas9载体及其构建方法 |
| JP2023503429A (ja) | 2019-11-22 | 2023-01-30 | アンスティチュ ナショナル ドゥ ラ サンテ エ ドゥ ラ ルシェルシュ メディカル | 白血病幹細胞を根絶することによる、急性骨髄性白血病の処置のための、アドレノメデュリン阻害剤 |
| WO2021108363A1 (en) | 2019-11-25 | 2021-06-03 | Regeneron Pharmaceuticals, Inc. | Crispr/cas-mediated upregulation of humanized ttr allele |
| AR120563A1 (es) | 2019-11-26 | 2022-02-23 | Novartis Ag | Receptores de antígeno quimérico cd19 y cd22 y sus usos |
| WO2021105368A1 (en) | 2019-11-27 | 2021-06-03 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Use of neuropilin antagonists for the treatment of endometriosis |
| WO2021108324A1 (en) | 2019-11-27 | 2021-06-03 | Technical University Of Denmark | Constructs, compositions and methods thereof having improved genome editing efficiency and specificity |
| IL293204A (en) | 2019-11-27 | 2022-07-01 | Promega Corp | Multipartite luciferase peptides and polypeptides |
| CN114945273A (zh) | 2019-11-29 | 2022-08-26 | 巴斯夫欧洲公司 | 增加植物对抗真菌感染的抗性 |
| AU2020398213A1 (en) | 2019-12-03 | 2022-06-16 | Beam Therapeutics Inc. | Synthetic guide RNA, compositions, methods, and uses thereof |
| CN114787365A (zh) | 2019-12-03 | 2022-07-22 | 巴斯夫欧洲公司 | 用于在植物中增强基因表达的调节性核酸分子 |
| KR20250033331A (ko) | 2019-12-10 | 2025-03-07 | 인스크립타 인코포레이티드 | 신규 mad 뉴클레아제 |
| US10704033B1 (en) | 2019-12-13 | 2020-07-07 | Inscripta, Inc. | Nucleic acid-guided nucleases |
| GB201918586D0 (en) | 2019-12-17 | 2020-01-29 | Patterson James | Engineered platelets for targeted delivery of a therapeutic agent |
| WO2021126886A1 (en) | 2019-12-18 | 2021-06-24 | Inscripta, Inc. | Cascade/dcas3 complementation assays for in vivo detection of nucleic acid-guided nuclease edited cells |
| EP4077699A1 (en) | 2019-12-19 | 2022-10-26 | Basf Se | Increasing space-time-yield, carbon-conversion-efficiency and carbon substrate flexibility in the production of fine chemicals |
| MX2022007714A (es) | 2019-12-20 | 2022-07-19 | Basf Se | Disminucion de la toxicidad de terpenos y aumento de la posible produccion en microorganismos. |
| IL271656A (en) | 2019-12-22 | 2021-06-30 | Yeda Res & Dev | System and methods for identifying cells that have undergone genome editing |
| EP3842452A1 (en) | 2019-12-26 | 2021-06-30 | Universitat Autònoma de Barcelona | Scaffold proteins and therapeutic nanoconjugates based on nidogen |
| CN114901813A (zh) | 2019-12-30 | 2022-08-12 | 博雅缉因(北京)生物科技有限公司 | 一种靶向t细胞淋巴瘤细胞的通用型car-t及其制备方法和应用 |
| WO2021136415A1 (zh) | 2019-12-30 | 2021-07-08 | 博雅辑因(北京)生物科技有限公司 | 一种纯化ucart细胞的方法与应用 |
| US11859172B2 (en) | 2020-01-02 | 2024-01-02 | The Trustees Of Columbia University In The City Of New York | Programmable and portable CRISPR-Cas transcriptional activation in bacteria |
| CN110982820A (zh) * | 2020-01-03 | 2020-04-10 | 云南中烟工业有限责任公司 | 一种烟草单倍体的基因编辑方法 |
| US10689669B1 (en) | 2020-01-11 | 2020-06-23 | Inscripta, Inc. | Automated multi-module cell processing methods, instruments, and systems |
| WO2021144426A1 (en) | 2020-01-17 | 2021-07-22 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods and compositions for treating melanoma |
| CA3168604A1 (en) | 2020-01-21 | 2021-07-29 | Limagrain Europe | Wheat haploid inducer plant and uses |
| WO2021151085A2 (en) * | 2020-01-24 | 2021-07-29 | The General Hospital Corporation | Crispr-cas enzymes with enhanced on-target activity |
| WO2021151073A2 (en) | 2020-01-24 | 2021-07-29 | The General Hospital Corporation | Unconstrained genome targeting with near-pamless engineered crispr-cas9 variants |
| US11225674B2 (en) | 2020-01-27 | 2022-01-18 | Inscripta, Inc. | Electroporation modules and instrumentation |
| CN115175559A (zh) | 2020-01-28 | 2022-10-11 | 瑞泽恩制药公司 | 包含人源化pnpla3基因座的非人动物及其使用方法 |
| ES2981209T3 (es) | 2020-01-29 | 2024-10-07 | Sumitomo Chemical Co | Proceso de preparación de oligómero de ácido nucleico |
| CN115003682A (zh) | 2020-01-29 | 2022-09-02 | 住友化学株式会社 | 核酸寡聚物的制造方法 |
| WO2021158883A1 (en) | 2020-02-07 | 2021-08-12 | Regeneron Pharmaceuticals, Inc. | Non-human animals comprising a humanized klkb1 locus and methods of use |
| EP4107273A1 (en) | 2020-02-21 | 2022-12-28 | Limagrain Europe | Prime editing technology for plant genome engineering |
| US20230183377A1 (en) | 2020-02-26 | 2023-06-15 | Sorrento Therapeutics, Inc. | Activatable antigen binding proteins with universal masking moieties |
| WO2021178556A1 (en) | 2020-03-04 | 2021-09-10 | Regeneron Pharmaceuticals, Inc. | Methods and compositions for sensitization of tumor cells to immune therapy |
| EP4118207A1 (en) | 2020-03-11 | 2023-01-18 | Omega Therapeutics, Inc. | Compositions and methods for modulating forkhead box p3 (foxp3) gene expression |
| EP4119166A4 (en) | 2020-03-12 | 2024-06-05 | Institute for Basic Science | COMPOSITION FOR INDUCING APOPTOSIS OF CELLS HAVING GENOMIC SEQUENCE VARIATION AND METHOD FOR INDUCING APOPTOSIS OF CELLS USING THE COMPOSITION |
| EP4121522A4 (en) | 2020-03-19 | 2024-06-19 | Intellia Therapeutics, Inc. | METHODS AND COMPOSITIONS FOR DIRECTED GENOMIC EDITING |
| CN115916823A (zh) | 2020-03-20 | 2023-04-04 | 法国国家健康和医学研究院 | 人cd45rc特异的嵌合抗原受体及其用途 |
| EP4125348A1 (en) | 2020-03-23 | 2023-02-08 | Regeneron Pharmaceuticals, Inc. | Non-human animals comprising a humanized ttr locus comprising a v30m mutation and methods of use |
| WO2021193865A1 (ja) | 2020-03-26 | 2021-09-30 | 国立研究開発法人農業・食品産業技術総合研究機構 | 温度感受性雄性不稔植物の製造方法 |
| EP4130018A4 (en) | 2020-03-27 | 2024-04-17 | Sumitomo Chemical Company, Limited | Method for producing nucleic acid oligomer |
| WO2021202938A1 (en) | 2020-04-03 | 2021-10-07 | Creyon Bio, Inc. | Oligonucleotide-based machine learning |
| JP2023522848A (ja) | 2020-04-08 | 2023-06-01 | アストラゼネカ・アクチエボラーグ | 改善された部位特異的改変のための組成物及び方法 |
| US20230132275A1 (en) | 2020-04-08 | 2023-04-27 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Use of cdon inhibitors for the treatment of endothelial dysfunction |
| US20210332388A1 (en) | 2020-04-24 | 2021-10-28 | Inscripta, Inc. | Compositions, methods, modules and instruments for automated nucleic acid-guided nuclease editing in mammalian cells |
| WO2021217083A1 (en) | 2020-04-24 | 2021-10-28 | Sorrento Therapeutics, Inc. | Memory dimeric antigen receptors |
| TW202208626A (zh) * | 2020-04-24 | 2022-03-01 | 美商生命編輯公司 | Rna引導核酸酶及其活性片段與變體,以及使用方法 |
| JP2023522784A (ja) | 2020-04-27 | 2023-05-31 | ノバルティス アーゲー | 眼細胞療法のための方法及び組成物 |
| WO2021226141A1 (en) | 2020-05-04 | 2021-11-11 | Saliogen Therapeutics, Inc. | Transposition-based therapies |
| AU2021268263A1 (en) | 2020-05-05 | 2022-10-27 | Genus Plc | Methods for improving the health of porcine species by targeted inactivation of CD163 |
| WO2021224633A1 (en) | 2020-05-06 | 2021-11-11 | Orchard Therapeutics (Europe) Limited | Treatment for neurodegenerative diseases |
| WO2021224401A1 (en) | 2020-05-07 | 2021-11-11 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods and compositions for determining a reference range of β-galactose exposure platelet |
| JP2023525304A (ja) | 2020-05-08 | 2023-06-15 | ザ ブロード インスティテュート,インコーポレーテッド | 標的二本鎖ヌクレオチド配列の両鎖同時編集のための方法および組成物 |
| WO2021228944A1 (en) | 2020-05-13 | 2021-11-18 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Base editing approaches for the treatment of betahemoglobinopathies |
| WO2021230385A1 (en) | 2020-05-15 | 2021-11-18 | Astellas Pharma Inc. | Method for treating muscular dystrophy by targeting utrophin gene |
| US11787841B2 (en) | 2020-05-19 | 2023-10-17 | Inscripta, Inc. | Rationally-designed mutations to the thrA gene for enhanced lysine production in E. coli |
| US11263022B2 (en) * | 2020-05-21 | 2022-03-01 | Microsoft Technology Licensing, Llc | Mechanism to turn on/off post-processing features in the device media foundation transform |
| EP4161583A1 (en) | 2020-06-05 | 2023-04-12 | Institut National de la Santé et de la Recherche Médicale (INSERM) | Methods and pharmaceutical compositions for treating ocular diseases |
| JP2023530234A (ja) | 2020-06-05 | 2023-07-14 | ザ・ブロード・インスティテュート・インコーポレイテッド | 新生物を治療するための組成物および方法 |
| US20230332104A1 (en) | 2020-06-11 | 2023-10-19 | Novartis Ag | Zbtb32 inhibitors and uses thereof |
| CN111748539B (zh) * | 2020-06-11 | 2021-10-22 | 中国农业科学院农产品加工研究所 | CRISPR/LpCas9基因编辑系统及其应用 |
| US20230218608A1 (en) | 2020-06-18 | 2023-07-13 | INSERM (Institut National de la Santé et de la Recherche Médicale) | New strategy for treating pancreatic cancer |
| BR112022026509A2 (pt) | 2020-06-23 | 2023-03-07 | Univ Colorado Regents | Métodos para diagnosticar patógenos respiratórios e prever resultados relacionados à covid-19 |
| EP4171215A2 (en) | 2020-06-26 | 2023-05-03 | Regeneron Pharmaceuticals, Inc. | Non-human animals comprising a humanized ace2 locus |
| WO2022009052A2 (en) | 2020-07-06 | 2022-01-13 | Janssen Biotech, Inc. | Prostate neoantigens and their uses |
| WO2022008597A1 (en) | 2020-07-08 | 2022-01-13 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods and pharmaceutical composition for the treatment of infectious diseases |
| EP4179080A4 (en) * | 2020-07-08 | 2024-07-17 | The Jackson Laboratory | TRANSGENIC MOUSE MODELS SUPPORTING INNATURE HUMAN IMMUNE FUNCTION |
| US20230248810A1 (en) | 2020-07-09 | 2023-08-10 | Modalis Therapeutics Corporation | Method for treating alzheimer's disease by targeting mapt gene |
| US20230235315A1 (en) | 2020-07-10 | 2023-07-27 | Horizon Discovery Limited | Method for producing genetically modified cells |
| AU2021204717A1 (en) | 2020-07-15 | 2022-02-03 | Seminis Vegetable Seeds, Inc. | Green Bean Plants with Improved Disease Resistance |
| WO2022015969A1 (en) | 2020-07-15 | 2022-01-20 | LifeEDIT Therapeutics, Inc. | Uracil stabilizing proteins and active fragments and variants thereof and methods of use |
| WO2022029080A1 (en) | 2020-08-03 | 2022-02-10 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Population of treg cells functionally committed to exert a regulatory activity and their use for adoptive therapy |
| WO2022032085A1 (en) | 2020-08-07 | 2022-02-10 | The Jackson Laboratory | Targeted sequence insertion compositions and methods |
| WO2022036180A1 (en) | 2020-08-13 | 2022-02-17 | Yale University | Compositions and methods for engineering and selection of car t cells with desired phenotypes |
| US20220049303A1 (en) | 2020-08-17 | 2022-02-17 | Readcoor, Llc | Methods and systems for spatial mapping of genetic variants |
| CA3186862A1 (en) | 2020-08-18 | 2022-02-24 | Pioneer Hi-Bred International, Inc. | Multiple disease resistance genes and genomic stacks thereof |
| PL4058474T3 (pl) | 2020-08-20 | 2024-09-23 | A2 Biotherapeutics, Inc. | Kompozycje i sposoby leczenia nowotworów egfr-dodatnich |
| EP4097486A4 (en) | 2020-08-20 | 2023-09-06 | A2 Biotherapeutics, Inc. | COMPOSITIONS AND METHODS OF TREATMENT OF POSITIVE CANCERS AT CEACAM |
| WO2022040454A1 (en) | 2020-08-20 | 2022-02-24 | A2 Biotherapeutics, Inc. | Compositions and methods for treating mesothelin positive cancers |
| WO2022046662A1 (en) * | 2020-08-24 | 2022-03-03 | Metagenomi Ip Technologies, Llc | Systems and methods for transposing cargo nucleotide sequences |
| US20240238344A1 (en) | 2020-08-28 | 2024-07-18 | Vor Biopharma Inc. | Compositions and methods for cd123 modification |
| JP2023540276A (ja) | 2020-08-28 | 2023-09-22 | ブイオーアール バイオファーマ インコーポレーテッド | Cll1改変のための組成物および方法 |
| CA3193424A1 (en) | 2020-08-31 | 2022-03-03 | Yale University | Compositions and methods for delivery of nucleic acids to cells |
| WO2022050413A1 (ja) | 2020-09-04 | 2022-03-10 | 国立大学法人神戸大学 | 小型化シチジンデアミナーゼを含む二本鎖dnaの改変用複合体 |
| IL301139A (en) | 2020-09-11 | 2023-05-01 | Lifeedit Therapeutics Inc | Dna modifying enzymes and active fragments and variants thereof and methods of use |
| WO2022056459A1 (en) | 2020-09-14 | 2022-03-17 | Vor Biopharma, Inc. | Compositions and methods for cd5 modification |
| US20230398219A1 (en) | 2020-09-14 | 2023-12-14 | Vor Biopharma Inc. | Compositions and methods for cd38 modification |
| US11299731B1 (en) | 2020-09-15 | 2022-04-12 | Inscripta, Inc. | CRISPR editing to embed nucleic acid landing pads into genomes of live cells |
| WO2022061115A1 (en) | 2020-09-18 | 2022-03-24 | Vor Biopharma Inc. | Compositions and methods for cd7 modification |
| AU2021343526A1 (en) | 2020-09-18 | 2023-05-25 | Celyntra Therapeutics Sa | Constructs and uses thereof for efficient and specific genome editing |
| JPWO2022064908A1 (ko) | 2020-09-24 | 2022-03-31 | ||
| WO2022066973A1 (en) | 2020-09-24 | 2022-03-31 | Fred Hutchinson Cancer Research Center | Immunotherapy targeting pbk or oip5 antigens |
| JP2023543218A (ja) * | 2020-09-24 | 2023-10-13 | フラッグシップ パイオニアリング イノベーションズ ブイ, インコーポレイテッド | 遺伝子発現を阻害するための組成物及び方法 |
| CN116724053A (zh) | 2020-09-24 | 2023-09-08 | 弗雷德哈钦森癌症中心 | 靶向sox2抗原的免疫治疗 |
| WO2022067240A1 (en) | 2020-09-28 | 2022-03-31 | Vor Biopharma, Inc. | Compositions and methods for cd6 modification |
| EP4222167A1 (en) | 2020-09-30 | 2023-08-09 | Nobell Foods, Inc. | Recombinant milk proteins and food compositions comprising the same |
| US10947552B1 (en) | 2020-09-30 | 2021-03-16 | Alpine Roads, Inc. | Recombinant fusion proteins for producing milk proteins in plants |
| EP4222264A1 (en) | 2020-09-30 | 2023-08-09 | CRISPR Therapeutics AG | Materials and methods for treatment of amyotrophic lateral sclerosis |
| US10894812B1 (en) | 2020-09-30 | 2021-01-19 | Alpine Roads, Inc. | Recombinant milk proteins |
| US20230364146A1 (en) | 2020-09-30 | 2023-11-16 | Vor Biopharma Inc. | Compositions and methods for cd30 gene modification |
| WO2022069756A1 (en) | 2020-10-02 | 2022-04-07 | Limagrain Europe | Crispr-mediated directed codon re-write |
| EP4226160A1 (en) | 2020-10-05 | 2023-08-16 | Institut National de la Santé et de la Recherche Médicale (INSERM) | Gdf3 as biomarker and biotarget in post-ischemic cardiac remodeling |
| EP4225914A1 (en) | 2020-10-06 | 2023-08-16 | Keygene N.V. | Targeted sequence addition |
| TW202222841A (zh) | 2020-10-06 | 2022-06-16 | 福瑞德哈金森腫瘤研究中心 | 用於治療表現mage-a1之疾病的組成物及方法 |
| EP4229199A1 (en) | 2020-10-13 | 2023-08-23 | Centre National De La Recherche Scientifique (Cnrs) | Targeted-antibacterial-plasmids combining conjugation and crispr /cas systems and uses thereof |
| US20220145293A1 (en) | 2020-10-21 | 2022-05-12 | Massachusetts Institute Of Technology | Systems, methods, and compositions for site-specific genetic engineering using programmable addition via site-specific targeting elements (paste) |
| WO2022084531A1 (en) | 2020-10-23 | 2022-04-28 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods and compositions for treating glioma |
| US20240000846A1 (en) | 2020-10-27 | 2024-01-04 | Vor Biopharma Inc. | Compositions and methods for treating hematopoietic malignancy |
| US20240344058A1 (en) | 2020-10-30 | 2024-10-17 | Vor Biopharma Inc. | Compositions and methods for bcma modification |
| CN112553195B (zh) * | 2020-11-05 | 2022-04-05 | 南方医科大学 | 一种用于CRISPR-Cas9定点突变编辑DNMT1基因的试剂及其应用 |
| EP4242237A4 (en) | 2020-11-06 | 2025-01-01 | Editforce, Inc. | FOKI NUCLEASE DOMAIN VARIANT |
| KR20220062951A (ko) | 2020-11-09 | 2022-05-17 | 삼성전자주식회사 | 전자 장치 및 그의 연락처를 관리하는 방법 |
| US11512297B2 (en) | 2020-11-09 | 2022-11-29 | Inscripta, Inc. | Affinity tag for recombination protein recruitment |
| JP2023550355A (ja) | 2020-11-13 | 2023-12-01 | ブイオーアール バイオファーマ インコーポレーテッド | キメラ抗原受容体を発現する遺伝子操作された細胞に関連する方法および組成物 |
| CN116635062A (zh) | 2020-11-13 | 2023-08-22 | 诺华股份有限公司 | 使用表达嵌合抗原受体(car)的细胞的组合疗法 |
| EP4244391A1 (en) | 2020-11-16 | 2023-09-20 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods and compositions for predicting and treating uveal melanoma |
| US20240011040A1 (en) | 2020-11-24 | 2024-01-11 | University Of Houston System | Salicylic acid-inducible gene expression compositions and systems for cells |
| WO2022112316A1 (en) | 2020-11-24 | 2022-06-02 | Keygene N.V. | Targeted enrichment using nanopore selective sequencing |
| US11459372B2 (en) | 2020-11-30 | 2022-10-04 | Crispr Therapeutics Ag | Gene-edited natural killer cells |
| EP4256052A1 (en) | 2020-12-02 | 2023-10-11 | Decibel Therapeutics, Inc. | Crispr sam biosensor cell lines and methods of use thereof |
| TW202237836A (zh) | 2020-12-03 | 2022-10-01 | 美商斯奎柏治療公司 | 工程化之第2類第v型crispr系統 |
| EP4259651A2 (en) | 2020-12-14 | 2023-10-18 | Fred Hutchinson Cancer Center | Compositions and methods for cellular immunotherapy |
| KR20230124682A (ko) | 2020-12-23 | 2023-08-25 | 플래그쉽 파이어니어링 이노베이션스 브이, 인크. | Rna를 봉입하는 아넬로바이러스 캡시드의 시험관 내어셈블리 |
| US11473060B2 (en) | 2020-12-30 | 2022-10-18 | Crispr Therapeutics Ag | Compositions and methods for differentiating stem cells into NK cells |
| TW202235615A (zh) | 2020-12-30 | 2022-09-16 | 美商英特利亞醫療公司 | 工程化之t細胞 |
| US11566230B2 (en) | 2020-12-31 | 2023-01-31 | Crispr Therapeutics Ag | Universal donor cells |
| CA3202219A1 (en) | 2020-12-31 | 2022-07-07 | Vor Biopharma Inc. | Compositions and methods for cd34 gene modification |
| AU2021415461A1 (en) | 2021-01-04 | 2023-08-17 | Inscripta, Inc. | Mad nucleases |
| EP4274893B1 (en) | 2021-01-05 | 2025-01-01 | Revvity Discovery Limited | Method for producing genetically modified cells |
| WO2022150269A1 (en) | 2021-01-07 | 2022-07-14 | Inscripta, Inc. | Mad nucleases |
| EP4277989A2 (en) | 2021-01-12 | 2023-11-22 | March Therapeutics, Inc. | Context-dependent, double-stranded dna-specific deaminases and uses thereof |
| AU2022216614A1 (en) | 2021-02-05 | 2023-02-23 | Christiana Care Gene Editing Institute, Inc. | Methods of and compositions for reducing gene expression and/or activity |
| US20240425885A1 (en) * | 2021-02-08 | 2024-12-26 | Emendobio Inc. | OMNI 90-99, 101, 104-110, 114, 116, 118-123, 125, 126, 128, 129, and 131-138 CRISPR NUCLEASE |
| CA3206576A1 (en) * | 2021-02-08 | 2022-08-11 | Lior IZHAR | Omni 103 crispr nuclease |
| AU2022221606A1 (en) | 2021-02-16 | 2023-08-24 | A2 Biotherapeutics, Inc. | Compositions and methods for treating her2 positive cancers |
| US11884924B2 (en) | 2021-02-16 | 2024-01-30 | Inscripta, Inc. | Dual strand nucleic acid-guided nickase editing |
| WO2022182801A1 (en) | 2021-02-25 | 2022-09-01 | Artisan Development Labs, Inc. | Compositions and methods for targeting, editing, or modifying genes |
| US20240052372A1 (en) | 2021-02-25 | 2024-02-15 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Allele-specific genome editing of the nr2e3 mutation g56r |
| US20240141399A1 (en) * | 2021-03-01 | 2024-05-02 | The Regents Of The University Of California | Methods for generating a crispr array |
| GB202103131D0 (en) | 2021-03-05 | 2021-04-21 | Biosystems Tech Limited | Method for preparation of research organisms |
| WO2022194908A1 (en) | 2021-03-17 | 2022-09-22 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods and compositions for treating melanoma |
| TW202300649A (zh) | 2021-03-22 | 2023-01-01 | 美商生命編輯治療學公司 | Dna修飾酶及活性片段及其變體及使用方法 |
| WO2022214632A1 (en) | 2021-04-07 | 2022-10-13 | Neoplants Sas | Compositions and methods for indoor air remediation |
| US20240182890A1 (en) | 2021-04-07 | 2024-06-06 | Astrazeneca Ab | Compositions and methods for site-specific modification |
| WO2022217086A1 (en) | 2021-04-09 | 2022-10-13 | Vor Biopharma Inc. | Photocleavable guide rnas and methods of use thereof |
| US20240382592A1 (en) | 2021-04-09 | 2024-11-21 | Institut National de la Santé et de la Recherche Médicale | Methods for the treatment of anaplastic large cell lymphoma |
| EP4322742A1 (en) | 2021-04-15 | 2024-02-21 | Keygene N.V. | Co-regeneration recalcitrant plants |
| JP2024513588A (ja) | 2021-04-15 | 2024-03-26 | キージーン ナムローゼ フェンノートシャップ | 遺伝性突然変異のための可動性エンドヌクレアーゼ |
| AU2022256513A1 (en) | 2021-04-16 | 2023-11-02 | Beam Therapeutics Inc. | Genetic modification of hepatocytes |
| AU2022261124A1 (en) | 2021-04-22 | 2023-10-05 | Dana-Farber Cancer Institute, Inc. | Compositions and methods for treating cancer |
| EP4326903A1 (en) | 2021-04-23 | 2024-02-28 | Inserm (Institut National De La Sante Et De La Recherche Medicale) | Methods and compositions for treating cell senescence accumulation related disease |
| US20240247285A1 (en) | 2021-05-10 | 2024-07-25 | Sqz Biotechnologies Company | Methods for delivering genome editing molecules to the nucleus or cytosol of a cell and uses thereof |
| US20240384246A1 (en) | 2021-05-27 | 2024-11-21 | Astrazeneca Ab | Ca s9 effector proteins with enhanced stability |
| WO2022251644A1 (en) | 2021-05-28 | 2022-12-01 | Lyell Immunopharma, Inc. | Nr4a3-deficient immune cells and uses thereof |
| WO2022254337A1 (en) | 2021-06-01 | 2022-12-08 | Novartis Ag | Cd19 and cd22 chimeric antigen receptors and uses thereof |
| US20250179481A1 (en) | 2021-06-01 | 2025-06-05 | Celyntra Therapeutics Sa | Compositions and methods for targeting, editing, or modifying genes |
| KR20240031238A (ko) | 2021-06-01 | 2024-03-07 | 아버 바이오테크놀로지스, 인크. | Crispr 뉴클레이스를 포함하는 유전자 편집 시스템 및 이의 용도 |
| US20230052243A1 (en) | 2021-06-02 | 2023-02-16 | Lyell Immunopharma, Inc. | Nr4a-deficient cells expressing c-jun and uses thereof |
| CA3221008A1 (en) | 2021-06-02 | 2022-12-08 | Brian CAFFERTY | Circular guide rnas for crispr/cas editing systems |
| WO2022261115A1 (en) | 2021-06-07 | 2022-12-15 | Yale University | Peptide nucleic acids for spatiotemporal control of crispr-cas binding |
| AU2022290278A1 (en) | 2021-06-11 | 2024-01-04 | LifeEDIT Therapeutics, Inc. | Rna polymerase iii promoters and methods of use |
| GB202108585D0 (en) | 2021-06-16 | 2021-07-28 | Rockend Ltd | Methods and compositions |
| EP4370676A2 (en) | 2021-06-18 | 2024-05-22 | Artisan Development Labs, Inc. | Compositions and methods for targeting, editing or modifying human genes |
| WO2022272293A1 (en) * | 2021-06-23 | 2022-12-29 | The Board Of Trustees Of The Leland Stanford Junior University | Compositions and methods for efficient retron production and genetic editing |
| US20230014010A1 (en) | 2021-06-23 | 2023-01-19 | Crispr Therapeutics Ag | Engineered cells with improved protection from natural killer cell killing |
| WO2023283585A2 (en) | 2021-07-06 | 2023-01-12 | Vor Biopharma Inc. | Inhibitor oligonucleotides and methods of use thereof |
| EP4367242A2 (en) | 2021-07-07 | 2024-05-15 | Omega Therapeutics, Inc. | Compositions and methods for modulating secreted frizzled receptor protein 1 (sfrp1) gene expression |
| US20240352452A1 (en) * | 2021-07-09 | 2024-10-24 | The Brigham And Women's Hospital, Inc. | Crispr-based protein barcoding and surface assembly |
| WO2023288281A2 (en) | 2021-07-15 | 2023-01-19 | Fred Hutchinson Cancer Center | Chimeric polypeptides |
| WO2023004409A1 (en) | 2021-07-23 | 2023-01-26 | Beam Therapeutics Inc. | Guide rnas for crispr/cas editing systems |
| US20240254483A1 (en) | 2021-07-30 | 2024-08-01 | Tune Therapeutics, Inc. | Compositions and methods for modulating expression of frataxin (fxn) |
| US20240252684A1 (en) | 2021-07-30 | 2024-08-01 | Tune Therapeutics, Inc. | Compositions and methods for modulating expression of methyl-cpg binding protein 2 (mecp2) |
| WO2023015182A1 (en) | 2021-08-02 | 2023-02-09 | Vor Biopharma Inc. | Compositions and methods for gene modification |
| EP4144841A1 (en) * | 2021-09-07 | 2023-03-08 | Bayer AG | Novel small rna programmable endonuclease systems with impoved pam specificity and uses thereof |
| US12344870B2 (en) | 2021-09-08 | 2025-07-01 | Arbor Biotechnologies, Inc. | Compositions comprising a CRISPR nuclease and uses thereof |
| WO2023039586A1 (en) | 2021-09-10 | 2023-03-16 | Agilent Technologies, Inc. | Guide rnas with chemical modification for prime editing |
| WO2023049742A2 (en) * | 2021-09-21 | 2023-03-30 | Scribe Therapeutics Inc. | Engineered casx repressor systems |
| WO2023049898A1 (en) | 2021-09-27 | 2023-03-30 | Monsanto Technology Llc | Compositions and methods for transformation of monocot seed excised embryo explants |
| WO2023049926A2 (en) | 2021-09-27 | 2023-03-30 | Vor Biopharma Inc. | Fusion polypeptides for genetic editing and methods of use thereof |
| WO2023052366A1 (en) | 2021-09-28 | 2023-04-06 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Base editing approaches for the treatment of beta-hemoglobinopathies |
| EP4410770A1 (en) | 2021-09-28 | 2024-08-07 | Sumitomo Chemical Company, Limited | Production method for purified dichloroacetic acid |
| CA3233097A1 (en) | 2021-09-30 | 2023-04-06 | Katherine Diane GRIBBLE | Compositions and methods for treating kcnq4-associated hearing loss |
| EP4408996A2 (en) | 2021-09-30 | 2024-08-07 | Astrazeneca AB | Use of inhibitors to increase efficiency of crispr/cas insertions |
| WO2023064813A2 (en) * | 2021-10-13 | 2023-04-20 | University Of Massachusetts | Modified guide rnas for neisseria meningitidis cas9 |
| MX2024003887A (es) | 2021-10-14 | 2024-07-09 | Arsenal Biosciences Inc | Células inmunitarias que tienen arnch coespresados y sistemas de compuerta lógica. |
| KR20240082391A (ko) | 2021-10-14 | 2024-06-10 | 론자 세일즈 아게 | 세포외 소포 생산을 위한 변형된 생산자 세포 |
| US12285497B2 (en) | 2021-10-15 | 2025-04-29 | Research Institute At Nationwide Children's Hospital | Self-complementary adeno-associated virus vector and its use in treatment of muscular dystrophy |
| WO2023064732A1 (en) | 2021-10-15 | 2023-04-20 | Georgia State University Research Foundation, Inc. | Delivery of therapeutic recombinant uricase using nanoparticles |
| AU2022368907A1 (en) | 2021-10-20 | 2024-05-02 | University Of Copenhagen | Rejuvenation treatment of age-related white matter loss |
| WO2023070043A1 (en) | 2021-10-20 | 2023-04-27 | Yale University | Compositions and methods for targeted editing and evolution of repetitive genetic elements |
| US20240407344A1 (en) | 2021-10-22 | 2024-12-12 | Tokyo Metropolitan Institute Of Medical Science | Neurodegenerative and amyotrophic model animal |
| JP2024542982A (ja) | 2021-10-26 | 2024-11-19 | リジェネロン・ファーマシューティカルズ・インコーポレイテッド | タウオパチーの治療様式としてのlemd2、lemd3、又はchmp7の過剰発現 |
| WO2023077012A1 (en) | 2021-10-27 | 2023-05-04 | Regeneron Pharmaceuticals, Inc. | Compositions and methods for expressing factor ix for hemophilia b therapy |
| WO2023077053A2 (en) | 2021-10-28 | 2023-05-04 | Regeneron Pharmaceuticals, Inc. | Crispr/cas-related methods and compositions for knocking out c5 |
| IL312452A (en) | 2021-11-01 | 2024-06-01 | Tome Biosciences Inc | A transformant has a single structure for the simultaneous transfer of a gene editing mechanism and a nucleic acid cargo |
| EP4427044A1 (en) | 2021-11-03 | 2024-09-11 | Institut National de la Santé et de la Recherche Médicale (INSERM) | Methods and compositions for treating triple negative breast cancer (tnbc) |
| EP4426832A1 (en) | 2021-11-03 | 2024-09-11 | The J. David Gladstone Institutes, A Testamentary Trust Established under The Will of J. David Gladstone | Precise genome editing using retrons |
| KR20240107104A (ko) | 2021-11-04 | 2024-07-08 | 리제너론 파마슈티칼스 인코포레이티드 | 변형된 cacng1 유전자좌를 포함하는 비인간 동물 |
| CA3236152A1 (en) | 2021-11-09 | 2023-05-19 | Vor Biopharma Inc. | Compositions and methods for erm2 modification |
| EP4433512A2 (en) | 2021-11-19 | 2024-09-25 | The Trustees of The University of Pennsylvania | Engineered pan-leukocyte antigen cd45 to facilitate car t cell therapy |
| GB202117314D0 (en) | 2021-11-30 | 2022-01-12 | Clarke David John | Cyclic nucleic acid fragmentation |
| WO2023099591A1 (en) | 2021-12-01 | 2023-06-08 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods for increasing fetal hemoglobin content by editing the +55-kb region of the erythroid-specific bcl11a enhancer |
| CN118632622A (zh) | 2021-12-08 | 2024-09-10 | 瑞泽恩制药公司 | 突变型肌纤蛋白疾病模型及其用途 |
| WO2023105000A1 (en) | 2021-12-09 | 2023-06-15 | Zygosity Limited | Vector |
| WO2023105244A1 (en) | 2021-12-10 | 2023-06-15 | Pig Improvement Company Uk Limited | Editing tmprss2/4 for disease resistance in livestock |
| US20230193310A1 (en) | 2021-12-10 | 2023-06-22 | Seminis Vegetabe Seeds, Inc. | Lettuce plants having resistance to downy mildew |
| GB202118058D0 (en) | 2021-12-14 | 2022-01-26 | Univ Warwick | Methods to increase yields in crops |
| US20230279442A1 (en) | 2021-12-15 | 2023-09-07 | Versitech Limited | Engineered cas9-nucleases and method of use thereof |
| WO2023111173A1 (en) | 2021-12-16 | 2023-06-22 | INSERM (Institut National de la Santé et de la Recherche Médicale) | An ezh2 degrader or inhibitor for use in the treatment of resistant acute myeloid leukemia |
| WO2023111225A1 (en) | 2021-12-17 | 2023-06-22 | Keygene N.V. | Double decapitation of plants |
| WO2023122506A1 (en) | 2021-12-20 | 2023-06-29 | Regeneron Pharmaceuticals, Inc. | Non-human animals comprising humanized ace2 and tmprss loci |
| EP4453199A1 (en) | 2021-12-21 | 2024-10-30 | Benson Hill, Inc. | Compositions and methods for modifying genomes |
| EP4452257A1 (en) | 2021-12-21 | 2024-10-30 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods and compositions for treating melanoma |
| US20250197854A1 (en) | 2021-12-21 | 2025-06-19 | Alia Therapeutics Srl | Type ii cas proteins and applications thereof |
| US20250057980A1 (en) | 2021-12-22 | 2025-02-20 | Tome Biosciences, Inc. | Co-Delivery of a Gene Editor Construct and a Donor Template |
| EP4453212A1 (en) | 2021-12-23 | 2024-10-30 | University of Massachusetts | Therapeutic treatment for fragile x-associated disorder |
| AU2022424002A1 (en) | 2021-12-29 | 2024-06-13 | Bristol-Myers Squibb Company | Generation of landing pad cell lines |
| EP4463548A1 (en) | 2022-01-14 | 2024-11-20 | Tune Therapeutics, Inc. | Compositions, systems, and methods for programming t cell phenotypes through targeted gene activation |
| US20250154503A1 (en) | 2022-01-14 | 2025-05-15 | Tune Therapeutics, Inc. | Compositions, systems, and methods for programming t cell phenotypes through targeted gene repression |
| WO2023141602A2 (en) | 2022-01-21 | 2023-07-27 | Renagade Therapeutics Management Inc. | Engineered retrons and methods of use |
| WO2023139557A1 (en) | 2022-01-24 | 2023-07-27 | LifeEDIT Therapeutics, Inc. | Rna-guided nucleases and active fragments and variants thereof and methods of use |
| US20250161492A1 (en) | 2022-01-25 | 2025-05-22 | Institut National de la Santé et de la Recherche Médicale | Base editing approaches for the treatment of beta-thalassemia |
| WO2023144235A1 (en) | 2022-01-27 | 2023-08-03 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods for monitoring and treating warburg effect in patients with pi3k-related disorders |
| US20250230412A1 (en) | 2022-02-01 | 2025-07-17 | President And Fellows Of Harvard College | Methods and compositions for treating cancer |
| KR20240135629A (ko) | 2022-02-02 | 2024-09-11 | 리제너론 파마슈티칼스 인코포레이티드 | 폼페병의 치료를 위한 항-TfR:GAA 및 항-CD63:GAA 삽입 |
| WO2023150798A1 (en) | 2022-02-07 | 2023-08-10 | Regeneron Pharmaceuticals, Inc. | Compositions and methods for defining optimal treatment timeframes in lysosomal disease |
| EP4223877A1 (en) * | 2022-02-08 | 2023-08-09 | Eberhard Karls Universität Tübingen Medizinische Fakultät | System and method for editing genomic dna to modulate splicing |
| EP4476245A1 (en) | 2022-02-11 | 2024-12-18 | Regeneron Pharmaceuticals, Inc. | Compositions and methods for screening 4r tau targeting agents |
| US20250152677A1 (en) | 2022-02-14 | 2025-05-15 | Institut National de la Santé et de la Recherche Médicale | Treatment of liver cancers by disrupting the beta-catenin/tcf-4 binding site located upstreatm of meg3 in the dlk1/dio3 locus |
| EP4479536A1 (en) | 2022-02-17 | 2024-12-25 | The Board Of Regents Of The University Of Texas System | Crispr/spcas9 variant and methods for enhanced correcton of duchenne muscular dystrophy mutations |
| EP4479143A1 (en) | 2022-02-18 | 2024-12-25 | Institut National de la Santé et de la Recherche Médicale (INSERM) | Use of tcr-deficient car-tregs in combination with anti-tcr complex monoclonal antibodies for inducing durable tolerance |
| AU2022201166B2 (en) * | 2022-02-21 | 2024-02-22 | Zhuhai Shu Tong Medical Technology Co., Ltd. | Type ii crispr/cas9 genome editing system and the application thereof |
| US12157886B2 (en) * | 2022-02-21 | 2024-12-03 | Zhuhai Shu Tong Medical Technology Co., Ltd. | Type II CRISPR/Cas9 genome editing system and the application thereof |
| EP4482955A2 (en) * | 2022-02-25 | 2025-01-01 | Duke University | Crispr-cas9 compositions and methods with a novel cas9 protein for genome editing and gene regulation |
| EP4482960A1 (en) | 2022-02-25 | 2025-01-01 | Vor Biopharma Inc. | Compositions and methods for homology-directed repair gene modification |
| EP4486890A1 (en) | 2022-03-01 | 2025-01-08 | CRISPR Therapeutics AG | Methods and compositions for treating angiopoietin-like 3 (angptl3) related conditions |
| WO2023167882A1 (en) | 2022-03-01 | 2023-09-07 | Artisan Development Labs, Inc. | Composition and methods for transgene insertion |
| US20250145655A1 (en) | 2022-03-23 | 2025-05-08 | Sumitomo Chemical Company, Limited | Method for producing nucleic acid oligomer |
| WO2023192872A1 (en) | 2022-03-28 | 2023-10-05 | Massachusetts Institute Of Technology | Rna scaffolded wireframe origami and methods thereof |
| JP2025511728A (ja) | 2022-04-04 | 2025-04-16 | ブイオーアール バイオファーマ インコーポレーテッド | エピトープを操作することを媒介するための組成物及び方法 |
| WO2023194359A1 (en) | 2022-04-04 | 2023-10-12 | Alia Therapeutics Srl | Compositions and methods for treatment of usher syndrome type 2a |
| WO2023205744A1 (en) | 2022-04-20 | 2023-10-26 | Tome Biosciences, Inc. | Programmable gene insertion compositions |
| CN114864002B (zh) * | 2022-04-28 | 2023-03-10 | 广西科学院 | 一种基于深度学习的转录因子结合位点识别方法 |
| JP2025514304A (ja) | 2022-04-29 | 2025-05-02 | リジェネロン・ファーマシューティカルズ・インコーポレイテッド | 遺伝子治療法のための組織特異的遺伝子外セーフハーバーの同定 |
| JP2025515030A (ja) | 2022-05-02 | 2025-05-13 | フォンダジオン テレソン イーティーエス | 遺伝子編集用の相同性非依存性標的組込み |
| WO2023215725A1 (en) | 2022-05-02 | 2023-11-09 | Fred Hutchinson Cancer Center | Compositions and methods for cellular immunotherapy |
| WO2023215831A1 (en) | 2022-05-04 | 2023-11-09 | Tome Biosciences, Inc. | Guide rna compositions for programmable gene insertion |
| AU2023269134A1 (en) | 2022-05-09 | 2024-12-12 | Regeneron Pharmaceuticals, Inc. | Vectors and methods for in vivo antibody production |
| EP4522647A1 (en) | 2022-05-10 | 2025-03-19 | Institut National de la Santé et de la Recherche Médicale | Base editing approaches for correcting the cd39 (cag>tag) mutation in patients suffering from beta-thalassemia |
| JP2025516462A (ja) * | 2022-05-13 | 2025-05-30 | エスアールアイ インターナショナル | 内因性遺伝子への転写因子のプログラム可能なリクルートメント |
| EP4525892A1 (en) | 2022-05-19 | 2025-03-26 | Lyell Immunopharma, Inc. | Polynucleotides targeting nr4a3 and uses thereof |
| EP4526884A2 (en) | 2022-05-20 | 2025-03-26 | Artisan Development Labs, Inc. | Systems and methods for assessing risk of genome editing events |
| WO2023225670A2 (en) | 2022-05-20 | 2023-11-23 | Tome Biosciences, Inc. | Ex vivo programmable gene insertion |
| EP4532765A2 (en) | 2022-05-25 | 2025-04-09 | Flagship Pioneering Innovations VII, LLC | Compositions and methods for modulation of immune responses |
| WO2023230578A2 (en) | 2022-05-25 | 2023-11-30 | Flagship Pioneering Innovations Vii, Llc | Compositions and methods for modulating circulating factors |
| WO2023230566A2 (en) | 2022-05-25 | 2023-11-30 | Flagship Pioneering Innovations Vii, Llc | Compositions and methods for modulating cytokines |
| WO2023230549A2 (en) | 2022-05-25 | 2023-11-30 | Flagship Pioneering Innovations Vii, Llc | Compositions and methods for modulation of tumor suppressors and oncogenes |
| EP4533088A2 (en) | 2022-05-25 | 2025-04-09 | Flagship Pioneering Innovations VII, LLC | Compositions and methods for modulating genetic drivers |
| WO2023235677A1 (en) | 2022-05-31 | 2023-12-07 | Regeneron Pharmaceuticals, Inc. | Animal model of tdp-43 proteinopathy |
| WO2023235725A2 (en) | 2022-05-31 | 2023-12-07 | Regeneron Pharmaceuticals, Inc. | Crispr-based therapeutics for c9orf72 repeat expansion disease |
| JP2025521154A (ja) | 2022-05-31 | 2025-07-08 | リジェネロン・ファーマシューティカルズ・インコーポレイテッド | C9orf72反復伸長疾患のためのcrispr干渉療法 |
| WO2023233339A1 (en) | 2022-06-01 | 2023-12-07 | Crispr Therapeutics Ag | Compositions and methods for differentiating stem cells into nk cells |
| WO2023233342A2 (en) | 2022-06-01 | 2023-12-07 | Crispr Therapeutics Ag | Gene-edited natural killer cells |
| WO2023250288A2 (en) | 2022-06-21 | 2023-12-28 | Seminis Vegetable Seeds, Inc. | Novel qtls conferring resistance to cucumber mosaic virus |
| KR20250044484A (ko) | 2022-06-24 | 2025-03-31 | 튠 쎄라퓨틱스, 인코포레이티드 | 표적 유전자 억제를 통해 저밀도 지단백질을 감소시키기 위한 조성물, 시스템, 및 방법 |
| EP4547821A1 (en) | 2022-06-29 | 2025-05-07 | Intellia Therapeutics, Inc. | Engineered t cells |
| GB202209518D0 (en) | 2022-06-29 | 2022-08-10 | Snipr Biome Aps | Treating & preventing E coli infections |
| JP7152094B1 (ja) | 2022-06-30 | 2022-10-12 | リージョナルフィッシュ株式会社 | tracrRNAユニット、及びゲノム編集方法 |
| WO2024006802A1 (en) | 2022-06-30 | 2024-01-04 | Pioneer Hi-Bred International, Inc. | Artificial intelligence-mediated methods and systems for genome editing |
| GB2621813A (en) | 2022-06-30 | 2024-02-28 | Univ Newcastle | Preventing disease recurrence in Mitochondrial replacement therapy |
| EP4299739A1 (en) | 2022-06-30 | 2024-01-03 | Inari Agriculture Technology, Inc. | Compositions, systems, and methods for genome editing |
| EP4299733A1 (en) | 2022-06-30 | 2024-01-03 | Inari Agriculture Technology, Inc. | Compositions, systems, and methods for genome editing |
| WO2024005863A1 (en) | 2022-06-30 | 2024-01-04 | Inari Agriculture Technology, Inc. | Compositions, systems, and methods for genome editing |
| WO2024005864A1 (en) | 2022-06-30 | 2024-01-04 | Inari Agriculture Technology, Inc. | Compositions, systems, and methods for genome editing |
| JP2025521677A (ja) | 2022-07-06 | 2025-07-10 | アンスティチュ ナショナル ドゥ ラ サンテ エ ドゥ ラ ルシェルシュ メディカル | 増殖性糸球体腎炎を処置するための方法 |
| WO2024015881A2 (en) | 2022-07-12 | 2024-01-18 | Tune Therapeutics, Inc. | Compositions, systems, and methods for targeted transcriptional activation |
| WO2024015925A2 (en) | 2022-07-13 | 2024-01-18 | Vor Biopharma Inc. | Compositions and methods for artificial protospacer adjacent motif (pam) generation |
| WO2024013514A2 (en) | 2022-07-15 | 2024-01-18 | Pig Improvement Company Uk Limited | Gene edited livestock animals having coronavirus resistance |
| CN120344660A (zh) | 2022-07-18 | 2025-07-18 | 雷纳嘉德医疗管理公司 | 基因编辑组分、系统和使用方法 |
| EP4558633A1 (en) | 2022-07-22 | 2025-05-28 | Institut National de la Santé et de la Recherche Médicale | Base editing approaches for correcting the ivs2-1 (g>a) mutation in patients suffering from beta-thalassemia |
| WO2024020587A2 (en) | 2022-07-22 | 2024-01-25 | Tome Biosciences, Inc. | Pleiopluripotent stem cell programmable gene insertion |
| KR20250042151A (ko) | 2022-07-22 | 2025-03-26 | 더 존스 홉킨스 유니버시티 | 덴드리머를 이용한 표적 세포 내 crispr/cas 시스템 전달 및 유전자 편집 |
| WO2024026313A1 (en) | 2022-07-25 | 2024-02-01 | The Regents Of The University Of California | Methods of producing and using avian embryonic stem cells and avian telencephalic organoids |
| WO2024023734A1 (en) | 2022-07-26 | 2024-02-01 | Bit Bio Limited | MULTI-gRNA GENOME EDITING |
| KR20250046255A (ko) | 2022-07-28 | 2025-04-02 | 스미또모 가가꾸 가부시끼가이샤 | 티오화 용액 |
| IL317261A (en) | 2022-07-29 | 2025-01-01 | Regeneron Pharma | Non-human animals containing a modified transferrin receptor locus |
| AU2023314808A1 (en) | 2022-07-29 | 2025-03-20 | Regeneron Pharmaceuticals, Inc. | Compositions and methods for transferrin receptor (tfr)-mediated delivery to the brain and muscle |
| WO2024028433A1 (en) | 2022-08-04 | 2024-02-08 | Institut National de la Santé et de la Recherche Médicale | Methods for the treatment of lymphoproliferative disorders |
| CN119562964A (zh) | 2022-08-05 | 2025-03-04 | 瑞泽恩制药公司 | tdp-43的抗聚集变体 |
| EP4569122A2 (en) | 2022-08-09 | 2025-06-18 | Pioneer Hi-Bred International, Inc. | Guide polynucleotide multiplexing |
| EP4569093A1 (en) | 2022-08-12 | 2025-06-18 | Life Edit Therapeutics, Inc. | Rna-guided nucleases and active fragments and variants thereof and methods of use |
| IL318767A (en) | 2022-08-19 | 2025-04-01 | Tune Therapeutics Inc | Compounds, systems and methods for regulating hepatitis B virus through targeted gene repression |
| CN120322549A (zh) | 2022-08-25 | 2025-07-15 | 生命编辑治疗股份有限公司 | 利用锁核酸对向导rna化学修饰以用于rna引导的核酸酶介导的基因编辑 |
| WO2024044723A1 (en) | 2022-08-25 | 2024-02-29 | Renagade Therapeutics Management Inc. | Engineered retrons and methods of use |
| WO2024042165A2 (en) | 2022-08-26 | 2024-02-29 | UCB Biopharma SRL | Novel rna-guided nucleases and nucleic acid targeting systems comprising such rna-guided nucleases |
| WO2024042168A1 (en) | 2022-08-26 | 2024-02-29 | UCB Biopharma SRL | Novel rna-guided nucleases and nucleic acid targeting systems comprising such rna-guided nucleases |
| EP4580658A1 (en) | 2022-08-31 | 2025-07-09 | Institut National de la Santé et de la Recherche Médicale | Method to generate more efficient car-t cells |
| WO2024047247A1 (en) | 2022-09-02 | 2024-03-07 | Institut National de la Santé et de la Recherche Médicale | Base editing approaches for the treatment of amyotrophic lateral sclerosis |
| EP4583905A1 (en) | 2022-09-06 | 2025-07-16 | Institut National de la Santé et de la Recherche Médicale | Novel dual split car-t cells for the treatment of cd38-positive hematological malignancies |
| WO2024056659A1 (en) | 2022-09-13 | 2024-03-21 | Institut National de la Santé et de la Recherche Médicale | Method for treating prostate cancer and other epithelial cancers |
| EP4587564A2 (en) | 2022-09-16 | 2025-07-23 | Alia Therapeutics Srl | Enqp type ii cas proteins and applications thereof |
| CN120239746A (zh) | 2022-09-19 | 2025-07-01 | 图恩疗法股份有限公司 | 用于调节t细胞功能的组合物、系统和方法 |
| IL319696A (en) * | 2022-09-19 | 2025-05-01 | Emendobio Inc | Biallelic silencing of CISH |
| WO2024064824A2 (en) | 2022-09-21 | 2024-03-28 | Yale University | Compositions and methods for identification of membrane targets for enhancement of nk cell therapy |
| WO2024064958A1 (en) | 2022-09-23 | 2024-03-28 | Lyell Immunopharma, Inc. | Methods for culturing nr4a-deficient cells |
| WO2024064952A1 (en) | 2022-09-23 | 2024-03-28 | Lyell Immunopharma, Inc. | Methods for culturing nr4a-deficient cells overexpressing c-jun |
| KR20250075694A (ko) | 2022-09-28 | 2025-05-28 | 리제너론 파마슈티칼스 인코포레이티드 | 세포 기반 요법을 강화하기 위한 항체 저항성 변형 수용체 |
| US20240224964A9 (en) | 2022-09-29 | 2024-07-11 | Regeneron Pharmaceuticals, Inc. | Correction of hepatosteatosis in humanized liver animals through restoration of il6/il6r/gp130 signaling in human hepatocytes |
| WO2024073751A1 (en) | 2022-09-29 | 2024-04-04 | Vor Biopharma Inc. | Methods and compositions for gene modification and enrichment |
| WO2024077174A1 (en) | 2022-10-05 | 2024-04-11 | Lyell Immunopharma, Inc. | Methods for culturing nr4a-deficient cells |
| AU2023361162A1 (en) | 2022-10-11 | 2025-05-29 | Yale University | Compositions and methods of using cell-penetrating antibodies |
| JP7353602B1 (ja) | 2022-10-14 | 2023-10-02 | 株式会社インプランタイノベーションズ | ゲノム編集方法およびゲノム編集用組成物 |
| WO2024083579A1 (en) | 2022-10-20 | 2024-04-25 | Basf Se | Regulatory nucleic acid molecules for enhancing gene expression in plants |
| WO2024084034A1 (en) | 2022-10-21 | 2024-04-25 | Institut National de la Santé et de la Recherche Médicale | Methods and pharmaceutical compositions for the treatment of osteoarthritis |
| JPWO2024089953A1 (ko) | 2022-10-27 | 2024-05-02 | ||
| CN115678903B (zh) * | 2022-11-03 | 2024-04-02 | 贵州大学 | 一种白背飞虱Ago1基因、合成dsRNA的方法及其应用 |
| WO2024098002A1 (en) | 2022-11-04 | 2024-05-10 | Regeneron Pharmaceuticals, Inc. | Calcium voltage-gated channel auxiliary subunit gamma 1 (cacng1) binding proteins and cacng1-mediated delivery to skeletal muscle |
| WO2024095245A2 (en) | 2022-11-04 | 2024-05-10 | LifeEDIT Therapeutics, Inc. | Evolved adenine deaminases and rna-guided nuclease fusion proteins with internal insertion sites and methods of use |
| WO2024102434A1 (en) | 2022-11-10 | 2024-05-16 | Senda Biosciences, Inc. | Rna compositions comprising lipid nanoparticles or lipid reconstructed natural messenger packs |
| WO2024107765A2 (en) | 2022-11-14 | 2024-05-23 | Regeneron Pharmaceuticals, Inc. | Compositions and methods for fibroblast growth factor receptor 3-mediated delivery to astrocytes |
| WO2024105162A1 (en) | 2022-11-16 | 2024-05-23 | Alia Therapeutics Srl | Type ii cas proteins and applications thereof |
| IL320124A (en) | 2022-11-16 | 2025-06-01 | Regeneron Pharma | Fusion proteins that include membrane-bound IL-12 with protease-cleavable ligands |
| WO2024105633A1 (en) | 2022-11-18 | 2024-05-23 | Kyoto Prefectural Public University Corporation | Compositions for mitophagy induction and uses thereof |
| WO2024118882A1 (en) | 2022-12-01 | 2024-06-06 | Genencor International Bv | Iterative multiplex genome engineering in microbial cells using a selection marker swapping system |
| WO2024118881A1 (en) | 2022-12-01 | 2024-06-06 | Genencor International Bv | Iterative muliplex genome engineering in microbial cells using a bidirectional selection marker system |
| WO2024118876A1 (en) | 2022-12-01 | 2024-06-06 | Genencor International Bv | Iterative multiplex genome engineering in microbial cells using a recombinant self-excisable selection marker system |
| AU2023406926A1 (en) | 2022-12-01 | 2025-06-26 | Yale University | Stimuli-responsive traceless engineering platform for intracellular payload delivery |
| WO2024123786A1 (en) | 2022-12-06 | 2024-06-13 | Pioneer Hi-Bred International, Inc. | Methods and compositions for co-delivery of t-dnas expressing multiple guide polynucleotides into plants |
| WO2024123789A1 (en) | 2022-12-07 | 2024-06-13 | Sanofi | Predicting indel frequencies |
| WO2024127370A1 (en) | 2022-12-16 | 2024-06-20 | LifeEDIT Therapeutics, Inc. | Guide rnas that target trac gene and methods of use |
| WO2024127369A1 (en) | 2022-12-16 | 2024-06-20 | LifeEDIT Therapeutics, Inc. | Guide rnas that target foxp3 gene and methods of use |
| AU2023408647A1 (en) | 2022-12-22 | 2025-06-12 | Keygene N.V. | Regeneration by protoplast callus grafting |
| WO2024133723A1 (en) | 2022-12-22 | 2024-06-27 | Institut National de la Santé et de la Recherche Médicale | Methods for decreasing therapeutic acquired resistance to chemotherapy and/or radiotherapy |
| WO2024138194A1 (en) | 2022-12-22 | 2024-06-27 | Tome Biosciences, Inc. | Platforms, compositions, and methods for in vivo programmable gene insertion |
| WO2024138189A2 (en) | 2022-12-22 | 2024-06-27 | Intellia Therapeutics, Inc. | Methods for analyzing nucleic acid cargos of lipid nucleic acid assemblies |
| WO2024143276A1 (ja) | 2022-12-26 | 2024-07-04 | 住友化学株式会社 | オリゴヌクレオチドの製造方法 |
| WO2024149810A2 (en) | 2023-01-11 | 2024-07-18 | Alia Therapeutics Srl | Type ii cas proteins and applications thereof |
| WO2024151213A2 (en) | 2023-01-12 | 2024-07-18 | National University Of Singapore | Blockade of cd8 expression and chimeric antigen receptors for immunotherapy of t-cell and nk-cell malignancies |
| WO2024159071A1 (en) | 2023-01-27 | 2024-08-02 | Regeneron Pharmaceuticals, Inc. | Modified rhabdovirus glycoproteins and uses thereof |
| WO2024163650A1 (en) | 2023-02-01 | 2024-08-08 | Regeneron Pharmaceuticals, Inc. | Animals comprising a modified klhdc7b locus |
| WO2024163683A2 (en) | 2023-02-01 | 2024-08-08 | Tune Therapeutics, Inc. | Systems, compositions, and methods for modulating expression of methyl-cpg binding protein 2 (mecp2) and x-inactive specific transcript (xist) |
| WO2024163678A2 (en) | 2023-02-01 | 2024-08-08 | Tune Therapeutics, Inc. | Fusion proteins and systems for targeted activation of frataxin (fxn) and related methods |
| WO2024163615A1 (en) | 2023-02-02 | 2024-08-08 | University Of Florida Research Foundation, Incorporated | Brain-derived neurotrophic factor-nano luciferase transgenic rodents and methods of use thereof |
| WO2024165484A1 (en) | 2023-02-06 | 2024-08-15 | Institut National de la Santé et de la Recherche Médicale | Enrichment of genetically modified hematopoietic stem cells through multiplex base editing |
| WO2024168097A2 (en) * | 2023-02-07 | 2024-08-15 | Applied Stemcell, Inc. | Integrase variants for gene insertion in human cell |
| WO2024168312A1 (en) | 2023-02-09 | 2024-08-15 | Vor Biopharma Inc. | Methods for treating hematopoietic malignancy |
| WO2024168348A1 (en) * | 2023-02-10 | 2024-08-15 | Tryptagenix, Inc. | Production of monoterpene indole alkaloid compounds in a heterologous host |
| WO2024173645A1 (en) | 2023-02-15 | 2024-08-22 | Arbor Biotechnologies, Inc. | Gene editing method for inhibiting aberrant splicing in stathmin 2 (stmn2) transcript |
| IT202300004443A1 (it) | 2023-03-09 | 2024-09-09 | Int Centre For Genetic Engineering And Biotechnology Icgeb | Sequenza codificante per alfa galattosidasi a umana per il trattamento della malattia di fabry |
| WO2024196921A1 (en) | 2023-03-20 | 2024-09-26 | Pioneer Hi-Bred International, Inc. | Cas polypeptides with altered pam recognition |
| WO2024196965A1 (en) | 2023-03-23 | 2024-09-26 | Carbon Biosciences, Inc. | Parvovirus compositions and related methods for gene therapy |
| WO2024197242A1 (en) | 2023-03-23 | 2024-09-26 | Carbon Biosciences, Inc. | Protoparvovirus compositions comprising a protoparvovirus variant vp1 capsid polypeptide and related methods |
| WO2024201368A1 (en) | 2023-03-29 | 2024-10-03 | Astrazeneca Ab | Use of inhibitors to increase efficiency of crispr/cas insertions |
| WO2024206911A2 (en) | 2023-03-30 | 2024-10-03 | Children's Hospital Medical Center | Clinical-grade organoids |
| WO2024206714A1 (en) * | 2023-03-31 | 2024-10-03 | Mammoth Biosciences, Inc. | Engineered effector proteins, compositions, systems and methods of use thereof |
| WO2024211287A1 (en) | 2023-04-03 | 2024-10-10 | Seagen Inc. | Production cell lines with targeted integration sites |
| WO2024211887A1 (en) * | 2023-04-07 | 2024-10-10 | Genentech, Inc. | Modified guide rnas |
| EP4445723A1 (en) | 2023-04-14 | 2024-10-16 | Seminis Vegetable Seeds, Inc. | Methods and compositions for peronospora resistance in spinach |
| WO2024220135A1 (en) | 2023-04-17 | 2024-10-24 | University Of Massachusetts | Prime editing systems having pegrna with reduced auto-inhibitory interaction |
| IT202300007968A1 (it) | 2023-04-21 | 2024-10-21 | Fond Telethon Ets | Metodi di editing genomico e costrutti |
| WO2024218295A1 (en) | 2023-04-21 | 2024-10-24 | Vib Vzw | Allelic combinations in crops for yield increase |
| WO2024226499A1 (en) | 2023-04-24 | 2024-10-31 | The Broad Institute, Inc. | Compositions and methods for modifying fertility |
| WO2024227131A1 (en) * | 2023-04-27 | 2024-10-31 | Rensselaer Polytechnic Institute | Recombinant enzyme for the accurate insertion of dna sequences in eukaryotic cells |
| WO2024226156A1 (en) | 2023-04-27 | 2024-10-31 | University Of Massachusetts | Cas-embedded cytidine deaminase ribonucleoprotein complexes having improved base editing specificity and efficiency |
| WO2024233328A1 (en) * | 2023-05-05 | 2024-11-14 | Insmed Incorporated | Nannochloropsis-producing viruses and methods and compositions for making the same |
| WO2024234006A1 (en) | 2023-05-11 | 2024-11-14 | Tome Biosciences, Inc. | Systems, compositions, and methods for targeting liver sinusodial endothelial cells (lsecs) |
| US20240417707A1 (en) * | 2023-05-11 | 2024-12-19 | University Of Massachusetts | Compositions and methods for improved genome editing with nme2cas9 and nme2-smucas9 variants |
| WO2024235991A1 (en) | 2023-05-15 | 2024-11-21 | UCB Biopharma SRL | Rna-guided nucleases and nucleic acid targeting systems comprising such rna-guided nucleases |
| WO2024236336A1 (en) | 2023-05-17 | 2024-11-21 | Institut National de la Santé et de la Recherche Médicale | Lipc variant in the treatment of hypercholesterolemia and atherosclerotic cardiovascular disease |
| WO2024238977A2 (en) | 2023-05-18 | 2024-11-21 | Children's Hospital Medical Center | Liver organoids with intrahepatic sympathetic nerves, and methods of use thereof |
| WO2024243438A2 (en) | 2023-05-23 | 2024-11-28 | Omega Therapeutics, Inc. | Compositions and methods for reducing cxcl9, cxcl10, and cxcl11 gene expression |
| WO2024245951A1 (en) | 2023-05-26 | 2024-12-05 | Institut National de la Santé et de la Recherche Médicale | Combination of slc8a1 inhibitor and mitochondria-targeted antioxidant for treating melanoma |
| WO2024246162A1 (en) | 2023-05-30 | 2024-12-05 | Institut National de la Santé et de la Recherche Médicale | Method and pharmaceutical composition for use in the treatment of focal cortical dysplasia |
| WO2024254376A1 (en) | 2023-06-08 | 2024-12-12 | Regeneron Pharmaceuticals, Inc. | Animal model with rapid onset of alzheimer's amyloid beta plaque pathology |
| WO2024259135A1 (en) | 2023-06-13 | 2024-12-19 | Intellia Therapeutics, Inc. | Assays for analysis of ribonucleic acid (rna) molecules |
| WO2024256635A1 (en) | 2023-06-15 | 2024-12-19 | Institut National de la Santé et de la Recherche Médicale | Dpm1 inhibitor for treating cancer |
| WO2024259309A1 (en) | 2023-06-15 | 2024-12-19 | Regeneron Pharmaceuticals, Inc. | Gene therapy for hearing disorders |
| CN118475621A (zh) | 2023-06-19 | 2024-08-09 | 杭州恩和生物科技有限公司 | 生产7-脱氢胆固醇的融合多肽及其使用方法 |
| WO2024261302A1 (en) | 2023-06-22 | 2024-12-26 | Institut National de la Santé et de la Recherche Médicale | Nlrp3 inhibitors, pak1/2 inhibitors and/or caspase 1 inhibitors for use in the treatment of rac2 monogenic disorders |
| WO2024263961A2 (en) | 2023-06-23 | 2024-12-26 | Children's Hospital Medical Center | Methods of matrix-free suspension culture |
| WO2024261323A1 (en) | 2023-06-23 | 2024-12-26 | Astrazeneca Ab | Molecular switches |
| WO2025003344A1 (en) | 2023-06-28 | 2025-01-02 | Alia Therapeutics Srl | Type ii cas proteins and applications thereof |
| US20250002946A1 (en) | 2023-06-30 | 2025-01-02 | Regeneron Pharmaceuticals, Inc. | Methods And Compositions For Increasing Homology-Directed Repair |
| WO2025012316A2 (en) | 2023-07-10 | 2025-01-16 | Universiteit Gent | Method of genome-editing |
| WO2025017030A1 (en) | 2023-07-17 | 2025-01-23 | Institut National de la Santé et de la Recherche Médicale | Prime editing of the -200 region in the hbg1 and/or hbg2 promoter for increasing fetal hemoglobin content in a eukaryotic cell |
| WO2025017033A1 (en) | 2023-07-17 | 2025-01-23 | Institut National de la Santé et de la Recherche Médicale | Prime editing of the -115 region in the hbg1 and/or hbg2 promoter for increasing fetal hemoglobin content in a eukaryotic cell |
| WO2025021839A1 (en) | 2023-07-25 | 2025-01-30 | Institut National de la Santé et de la Recherche Médicale | Method to treat metabolic disorders |
| WO2025022367A2 (en) | 2023-07-27 | 2025-01-30 | Life Edit Therapeutics, Inc. | Rna-guided nucleases and active fragments and variants thereof and methods of use |
| TW202513801A (zh) | 2023-07-28 | 2025-04-01 | 美商雷傑納榮製藥公司 | 用於治療龐貝氏症之抗TfR:GAA及抗CD63:GAA插入 |
| WO2025029654A2 (en) | 2023-07-28 | 2025-02-06 | Regeneron Pharmaceuticals, Inc. | Use of bgh-sv40l tandem polya to enhance transgene expression during unidirectional gene insertion |
| US20250049896A1 (en) | 2023-07-28 | 2025-02-13 | Regeneron Pharmaceuticals, Inc. | Anti-tfr:acid sphingomyelinase for treatment of acid sphingomyelinase deficiency |
| WO2025029840A1 (en) | 2023-07-31 | 2025-02-06 | Tune Therapeutics, Inc. | Compositions and methods for multiplexed activation and repression of t cell gene expression |
| WO2025029835A1 (en) | 2023-07-31 | 2025-02-06 | Tune Therapeutics, Inc. | Compositions and methods for modulating il-2 gene expression |
| WO2025027166A1 (en) | 2023-08-01 | 2025-02-06 | Basf Plant Science Company Gmbh | Increased resistance by expression of msbp1 protein |
| WO2025027165A1 (en) | 2023-08-01 | 2025-02-06 | Basf Plant Science Company Gmbh | Increased resistance by expression of an ics protein |
| WO2025030010A1 (en) | 2023-08-01 | 2025-02-06 | Vor Biopharma Inc. | Compositions comprising genetically engineered hematopoietic stem cells and methods of use thereof |
| WO2025032112A1 (en) | 2023-08-08 | 2025-02-13 | Institut National de la Santé et de la Recherche Médicale | Methods for the treatment of type 2-mediated diseases |
| WO2025038494A1 (en) | 2023-08-11 | 2025-02-20 | Tune Therapeutics, Inc. | Compositions, systems, and methods for lymphoid cell differentiation using targeted gene activation |
| WO2025038750A2 (en) | 2023-08-14 | 2025-02-20 | President And Fellows Of Harvard College | Methods and compositions for treating cancer |
| EP4512403A1 (en) | 2023-08-22 | 2025-02-26 | Friedrich-Schiller-Universität Jena | Neuropeptide b and w-receptor as a target for treating mood disorders and/or chronic stress |
| WO2025049524A1 (en) | 2023-08-28 | 2025-03-06 | Regeneron Pharmaceuticals, Inc. | Cxcr4 antibody-resistant modified receptors |
| WO2025050069A1 (en) | 2023-09-01 | 2025-03-06 | Tome Biosciences, Inc. | Programmable gene insertion using engineered integration enzymes |
| WO2025049959A2 (en) | 2023-09-01 | 2025-03-06 | Renagade Therapeutics Management Inc. | Gene editing systems, compositions, and methods for treatment of vexas syndrome |
| WO2025059073A1 (en) | 2023-09-11 | 2025-03-20 | Tune Therapeutics, Inc. | Epigenetic editing methods and systems for differentiating stem cells |
| WO2025059184A1 (en) | 2023-09-12 | 2025-03-20 | Pioneer Hi-Bred International, Inc. | Methods and compositions for generating genome-edited paternal doubled haploids |
| WO2025059215A1 (en) | 2023-09-12 | 2025-03-20 | Aadigen, Llc | Methods and compositions for treating or preventing cancer |
| WO2025064408A1 (en) | 2023-09-18 | 2025-03-27 | The Broad Institute, Inc. | Compositions and methods for treating cardiovascular disease |
| GB202314578D0 (en) | 2023-09-22 | 2023-11-08 | Univ Manchester | Methods of producing homoplasmic modified plants or parts thereof |
| WO2025072803A1 (en) | 2023-09-29 | 2025-04-03 | Children's Hospital Medical Center | Ntrk2 signaling-mediated alveolar capillary injury and repair |
| WO2025076141A1 (en) | 2023-10-03 | 2025-04-10 | Inari Agriculture Technology, Inc. | Viral delivery of grna to the scion |
| WO2025076306A1 (en) | 2023-10-06 | 2025-04-10 | University Of Massachusetts | Prime editors having improved prime editing efficiency |
| WO2025078851A1 (en) | 2023-10-11 | 2025-04-17 | Institut National De La Sante Et De La Recherche Medicale (Inserm) | Methods of treating cognitive deficit |
| WO2025081123A1 (en) | 2023-10-12 | 2025-04-17 | Fred Hutchinson Cancer Center | Methods and compositions for improving t cell immunotherapy |
| WO2025081042A1 (en) | 2023-10-12 | 2025-04-17 | Renagade Therapeutics Management Inc. | Nickase-retron template-based precision editing system and methods of use |
| WO2025078632A1 (en) | 2023-10-12 | 2025-04-17 | Institut National de la Santé et de la Recherche Médicale | Methods of prognosis and treatment of patients suffering from cancer |
| WO2025083169A1 (en) | 2023-10-17 | 2025-04-24 | Tenpoint Therapeutics Limited | Combination of a vegf inhibitor and a complement pathway inhibitor for treating ocular disorders |
| WO2025090427A1 (en) | 2023-10-23 | 2025-05-01 | University Of Rochester | Glial-targeted relief of hyperexcitability in neurodegenerative diseases |
| WO2025096638A2 (en) | 2023-10-30 | 2025-05-08 | Turnstone Biologics Corp. | Genetically modified tumor infilitrating lymphocytes and methods of producing and using the same |
| WO2025101820A1 (en) | 2023-11-08 | 2025-05-15 | Fred Hutchinson Cancer Center | Compositions and methods for cellular immunotherapy |
| US20250161347A1 (en) | 2023-11-22 | 2025-05-22 | Flagship Pioneering Innovations Vii, Llc | Methods and compositions for treating non-alcoholic fatty liver disease |
| WO2025122754A1 (en) | 2023-12-07 | 2025-06-12 | Regeneron Pharmaceuticals, Inc. | Gaa knockout non-human animals |
| WO2025132479A1 (en) | 2023-12-18 | 2025-06-26 | Institut National de la Santé et de la Recherche Médicale | Flt3 inhibitor for modulating macrophages polarization |
| WO2025137439A2 (en) | 2023-12-20 | 2025-06-26 | Intellia Therapeutics, Inc. | Engineered t cells |
| CN118127022B (zh) * | 2024-04-30 | 2024-07-12 | 四川大学 | 变异链球菌环状RNA circcsbD及应用、其过表达菌株及构建方法和应用 |
Family Cites Families (212)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US106048A (en) | 1870-08-02 | Materials of different | ||
| DE1133825B (de) | 1960-06-15 | 1962-07-26 | Danfoss Ved Ing M Clausen | Elektromagnetisches Relais |
| US3687808A (en) | 1969-08-14 | 1972-08-29 | Univ Leland Stanford Junior | Synthetic polynucleotides |
| US4952496A (en) | 1984-03-30 | 1990-08-28 | Associated Universities, Inc. | Cloning and expression of the gene for bacteriophage T7 RNA polymerase |
| US5034506A (en) | 1985-03-15 | 1991-07-23 | Anti-Gene Development Group | Uncharged morpholino-based polymers having achiral intersubunit linkages |
| WO1988008450A1 (en) | 1987-05-01 | 1988-11-03 | Birdwell Finlayson | Gene therapy for metabolite disorders |
| US5350689A (en) | 1987-05-20 | 1994-09-27 | Ciba-Geigy Corporation | Zea mays plants and transgenic Zea mays plants regenerated from protoplasts or protoplast-derived cells |
| US5585362A (en) | 1989-08-22 | 1996-12-17 | The Regents Of The University Of Michigan | Adenovirus vectors for gene therapy |
| US5451513A (en) | 1990-05-01 | 1995-09-19 | The State University of New Jersey Rutgers | Method for stably transforming plastids of multicellular plants |
| US5767367A (en) | 1990-06-23 | 1998-06-16 | Hoechst Aktiengesellschaft | Zea mays (L.) with capability of long term, highly efficient plant regeneration including fertile transgenic maize plants having a heterologous gene, and their preparation |
| US5602240A (en) | 1990-07-27 | 1997-02-11 | Ciba Geigy Ag. | Backbone modified oligonucleotide analogs |
| US5489677A (en) | 1990-07-27 | 1996-02-06 | Isis Pharmaceuticals, Inc. | Oligonucleoside linkages containing adjacent oxygen and nitrogen atoms |
| JPH06505186A (ja) | 1991-02-11 | 1994-06-16 | オマーヤ,アユブ ケー. | 脊髄液駆動式人工器官 |
| US5222982A (en) | 1991-02-11 | 1993-06-29 | Ommaya Ayub K | Spinal fluid driven artificial organ |
| US5539082A (en) | 1993-04-26 | 1996-07-23 | Nielsen; Peter E. | Peptide nucleic acids |
| US5719262A (en) | 1993-11-22 | 1998-02-17 | Buchardt, Deceased; Ole | Peptide nucleic acids having amino acid side chains |
| US5714331A (en) | 1991-05-24 | 1998-02-03 | Buchardt, Deceased; Ole | Peptide nucleic acids having enhanced binding affinity, sequence specificity and solubility |
| JP3534749B2 (ja) | 1991-08-20 | 2004-06-07 | アメリカ合衆国 | アデノウイルスが介在する胃腸管への遺伝子の輸送 |
| US7150982B2 (en) | 1991-09-09 | 2006-12-19 | Third Wave Technologies, Inc. | RNA detection assays |
| US5252479A (en) | 1991-11-08 | 1993-10-12 | Research Corporation Technologies, Inc. | Safe vector for gene therapy |
| FR2688514A1 (fr) | 1992-03-16 | 1993-09-17 | Centre Nat Rech Scient | Adenovirus recombinants defectifs exprimant des cytokines et medicaments antitumoraux les contenant. |
| US7153684B1 (en) | 1992-10-08 | 2006-12-26 | Vanderbilt University | Pluripotential embryonic stem cells and methods of making same |
| JPH08503855A (ja) | 1992-12-03 | 1996-04-30 | ジェンザイム・コーポレイション | 嚢胞性線維症に対する遺伝子治療 |
| EP0705344B8 (en) | 1993-06-24 | 2006-05-10 | Advec Inc. | Adenovirus vectors for gene therapy |
| ES2328585T3 (es) | 1993-10-25 | 2009-11-16 | Canji, Inc. | Vector de adenovirus recombinante y metodo de utilizacion. |
| US5576198A (en) | 1993-12-14 | 1996-11-19 | Calgene, Inc. | Controlled expression of transgenic constructs in plant plastids |
| US5545818A (en) | 1994-03-11 | 1996-08-13 | Calgene Inc. | Expression of Bacillus thuringiensis cry proteins in plant plastids |
| US5545817A (en) | 1994-03-11 | 1996-08-13 | Calgene, Inc. | Enhanced expression in a plant plastid |
| US5843780A (en) | 1995-01-20 | 1998-12-01 | Wisconsin Alumni Research Foundation | Primate embryonic stem cells |
| US6583336B1 (en) | 1995-08-30 | 2003-06-24 | Basf Plant Science Gmbh | Stimulation of homologous recombination in eukaryotic organisms or cells by recombination promoting enzymes |
| ES2222518T3 (es) | 1996-08-29 | 2005-02-01 | BAUSCH & LOMB INCORPORATED | Control de frecuencia y de potencia de dos bucles. |
| JP3756313B2 (ja) | 1997-03-07 | 2006-03-15 | 武 今西 | 新規ビシクロヌクレオシド及びオリゴヌクレオチド類縁体 |
| CA2303299C (en) | 1997-09-12 | 2016-02-23 | Exiqon A/S | Oligonucleotide analogues |
| JP3880795B2 (ja) | 1997-10-23 | 2007-02-14 | ジェロン・コーポレーション | フィーダー細胞を含まない培養物中で、霊長類由来始原幹細胞を増殖させるための方法 |
| US20040186071A1 (en) | 1998-04-13 | 2004-09-23 | Bennett C. Frank | Antisense modulation of CD40 expression |
| US20020182673A1 (en) | 1998-05-15 | 2002-12-05 | Genentech, Inc. | IL-17 homologous polypedies and therapeutic uses thereof |
| US7410798B2 (en) | 2001-01-10 | 2008-08-12 | Geron Corporation | Culture system for rapid expansion of human embryonic stem cells |
| US6667176B1 (en) | 2000-01-11 | 2003-12-23 | Geron Corporation | cDNA libraries reflecting gene expression during growth and differentiation of human pluripotent stem cells |
| US7078387B1 (en) | 1998-12-28 | 2006-07-18 | Arch Development Corp. | Efficient and stable in vivo gene transfer to cardiomyocytes using recombinant adeno-associated virus vectors |
| AU2982900A (en) | 1999-02-03 | 2000-08-25 | Children's Medical Center Corporation | Gene repair involving the induction of double-stranded dna cleavage at a chromosomal target site |
| US7229961B2 (en) | 1999-08-24 | 2007-06-12 | Cellgate, Inc. | Compositions and methods for enhancing drug delivery across and into ocular tissues |
| US6593292B1 (en) | 1999-08-24 | 2003-07-15 | Cellgate, Inc. | Compositions and methods for enhancing drug delivery across and into epithelial tissues |
| EP1083231A1 (en) | 1999-09-09 | 2001-03-14 | Introgene B.V. | Smooth muscle cell promoter and uses thereof |
| US7256286B2 (en) | 1999-11-30 | 2007-08-14 | The Board Of Trustees Of The Leland Stanford Junior University | Bryostatin analogues, synthetic methods and uses |
| US20020119570A1 (en) | 2000-09-25 | 2002-08-29 | Kyonggeun Yoon | Targeted gene correction by single-stranded oligodeoxynucleotides |
| MX357775B (es) | 2000-10-27 | 2018-07-20 | J Craig Venter Inst Inc | Acidos nucleicos y proteinas de los grupos a y b de estreptococos. |
| CA2437983C (en) | 2001-02-16 | 2011-10-25 | Cellgate, Inc. | Transporters comprising spaced arginine moieties |
| US7691995B2 (en) | 2001-07-12 | 2010-04-06 | University Of Massachusetts | In vivo production of small interfering RNAS that mediate gene silencing |
| US7169874B2 (en) | 2001-11-02 | 2007-01-30 | Bausch & Lomb Incorporated | High refractive index polymeric siloxysilane compositions |
| US20060253913A1 (en) | 2001-12-21 | 2006-11-09 | Yue-Jin Huang | Production of hSA-linked butyrylcholinesterases in transgenic mammals |
| BRPI0307383B1 (pt) | 2002-01-23 | 2019-12-31 | The Univ Of Utah Research Foundation | método de recombinação genética direcionada em célula de planta hospedeira |
| AU2003218382B2 (en) | 2002-03-21 | 2007-12-13 | Sangamo Therapeutics, Inc. | Methods and compositions for using zinc finger endonucleases to enhance homologous recombination |
| WO2003087993A2 (en) | 2002-04-09 | 2003-10-23 | Beattie Kenneth L | Oligonucleotide probes for genosensor chips |
| JP2006502748A (ja) | 2002-09-05 | 2006-01-26 | カリフォルニア インスティテュート オブ テクノロジー | 遺伝子ターゲッティングを誘発するキメラヌクレアーゼの使用方法 |
| WO2005070948A1 (en) | 2004-01-23 | 2005-08-04 | Intronn, Inc. | Correction of alpha-1-antitrypsin genetic defects using spliceosome mediated rna trans splicing |
| US7972854B2 (en) | 2004-02-05 | 2011-07-05 | Sangamo Biosciences, Inc. | Methods and compositions for targeted cleavage and recombination |
| US7919277B2 (en) | 2004-04-28 | 2011-04-05 | Danisco A/S | Detection and typing of bacterial strains |
| US7892224B2 (en) | 2005-06-01 | 2011-02-22 | Brainlab Ag | Inverse catheter planning |
| US7534819B2 (en) | 2005-06-10 | 2009-05-19 | University Of Washington | Compositions and methods for intracellular delivery of biotinylated cargo |
| KR20080033455A (ko) | 2005-07-26 | 2008-04-16 | 상가모 바이오사이언스 인코포레이티드 | 외래 핵산 서열의 표적화된 통합 및 발현 |
| US10022457B2 (en) | 2005-08-05 | 2018-07-17 | Gholam A. Peyman | Methods to regulate polarization and enhance function of cells |
| WO2007025091A2 (en) | 2005-08-24 | 2007-03-01 | Thomson Licensing | Method and apparatus for pop screen dialog for roving devices with terrestrial broadcast receiver |
| DK3284833T3 (da) * | 2005-08-26 | 2022-02-07 | Dupont Nutrition Biosci Aps | Anvendelse af crispr-associerede gener (cas) |
| US8278104B2 (en) | 2005-12-13 | 2012-10-02 | Kyoto University | Induced pluripotent stem cells produced with Oct3/4, Klf4 and Sox2 |
| AU2006325975B2 (en) | 2005-12-13 | 2011-12-08 | Kyoto University | Nuclear reprogramming factor |
| US20090227032A1 (en) | 2005-12-13 | 2009-09-10 | Kyoto University | Nuclear reprogramming factor and induced pluripotent stem cells |
| EP1994182B1 (en) | 2006-03-15 | 2019-05-29 | Siemens Healthcare Diagnostics Inc. | Degenerate nucleobase analogs |
| PL2019683T5 (pl) | 2006-04-25 | 2022-12-05 | The Regents Of The University Of California | Podawanie czynników wzrostu do leczenia zaburzeń OUN |
| ATE513053T1 (de) | 2006-05-10 | 2011-07-15 | Deinove Sa | Verfahren zur chromosomenmanipulation unter verwendung eines neuen dna-reparatursystems |
| PL2018441T3 (pl) | 2006-05-19 | 2012-03-30 | Dupont Nutrition Biosci Aps | Mikroorganizmy znakowane i sposoby znakowania |
| AU2007267874B2 (en) | 2006-05-25 | 2012-03-15 | Sangamo Therapeutics, Inc. | Methods and compositions for gene inactivation |
| AU2007258872A1 (en) | 2006-06-16 | 2007-12-21 | Danisco A/S | Bacterium |
| US8481272B2 (en) | 2006-08-04 | 2013-07-09 | Georgia State University Research Foundation, Inc. | Enzyme sensors, methods for preparing and using such sensors, and methods of detecting protease activity |
| WO2008060360A2 (en) | 2006-09-28 | 2008-05-22 | Surmodics, Inc. | Implantable medical device with apertures for delivery of bioactive agents |
| MX2009009071A (es) | 2007-03-02 | 2009-09-24 | Danisco | Cultivos con resistencia mejorada a fagos. |
| WO2008148304A1 (fr) | 2007-05-31 | 2008-12-11 | Xiamen University | Cible d'interférence arn pour traiter le sida |
| JP2008307007A (ja) | 2007-06-15 | 2008-12-25 | Bayer Schering Pharma Ag | 出生後のヒト組織由来未分化幹細胞から誘導したヒト多能性幹細胞 |
| WO2009073711A1 (en) * | 2007-12-04 | 2009-06-11 | Remedy Pharmaceuticals, Inc. | Improved formulations and methods for lyophilization and lyophilates provided thereby |
| US9683232B2 (en) | 2007-12-10 | 2017-06-20 | Kyoto University | Efficient method for nuclear reprogramming |
| EP2257250A2 (en) | 2008-01-29 | 2010-12-08 | Gilbert H. Kliman | Drug delivery devices, kits and methods therefor |
| WO2010011961A2 (en) * | 2008-07-25 | 2010-01-28 | University Of Georgia Research Foundation, Inc. | Prokaryotic rnai-like system and methods of use |
| JP5908725B2 (ja) | 2008-08-22 | 2016-04-26 | サンガモ バイオサイエンシーズ, インコーポレイテッド | 標的一本鎖開裂および標的組込みのための方法、並びに組成物 |
| MX2011002731A (es) | 2008-09-15 | 2011-04-26 | Childrens Medical Center | Modulacion de bcl11a para tratamiento de hemoglobinopatias. |
| US20100076057A1 (en) * | 2008-09-23 | 2010-03-25 | Northwestern University | TARGET DNA INTERFERENCE WITH crRNA |
| WO2010054108A2 (en) | 2008-11-06 | 2010-05-14 | University Of Georgia Research Foundation, Inc. | Cas6 polypeptides and methods of use |
| US10662227B2 (en) | 2008-11-07 | 2020-05-26 | Dupont Nutrition Biosciences Aps | Bifidobacteria CRISPR sequences |
| EP2367938B1 (en) | 2008-12-12 | 2014-06-11 | DuPont Nutrition Biosciences ApS | Genetic cluster of strains of streptococcus thermophilus having unique rheological properties for dairy fermentation |
| WO2010075424A2 (en) | 2008-12-22 | 2010-07-01 | The Regents Of University Of California | Compositions and methods for downregulating prokaryotic genes |
| GB0823658D0 (en) | 2008-12-30 | 2009-02-04 | Angiomed Ag | Stent delivery device |
| EP2206723A1 (en) | 2009-01-12 | 2010-07-14 | Bonas, Ulla | Modular DNA-binding domains |
| WO2010117646A1 (en) | 2009-04-09 | 2010-10-14 | Motorola, Inc. | Retransmission technique for a communication network |
| WO2010117464A1 (en) | 2009-04-09 | 2010-10-14 | Sangamo Biosciences, Inc. | Targeted integration into stem cells |
| SMT202000224T1 (it) | 2009-04-30 | 2020-05-08 | Ospedale San Raffaele Srl | Vettore genico |
| KR20120097483A (ko) | 2009-07-24 | 2012-09-04 | 시그마-알드리치 컴퍼니., 엘엘씨 | 게놈 편집을 위한 방법 |
| US20120192298A1 (en) | 2009-07-24 | 2012-07-26 | Sigma Aldrich Co. Llc | Method for genome editing |
| CA2769262C (en) | 2009-07-28 | 2019-04-30 | Sangamo Biosciences, Inc. | Methods and compositions for treating trinucleotide repeat disorders |
| US20110104787A1 (en) | 2009-11-05 | 2011-05-05 | President And Fellows Of Harvard College | Fusion Peptides That Bind to and Modify Target Nucleic Acid Sequences |
| US20110294114A1 (en) | 2009-12-04 | 2011-12-01 | Cincinnati Children's Hospital Medical Center | Optimization of determinants for successful genetic correction of diseases, mediated by hematopoietic stem cells |
| EP3456826B1 (en) * | 2009-12-10 | 2023-06-28 | Regents of the University of Minnesota | Tal effector-mediated dna modification |
| EP2660318A1 (en) | 2010-02-09 | 2013-11-06 | Sangamo BioSciences, Inc. | Targeted genomic modification with partially single-stranded donor molecules |
| US10087431B2 (en) | 2010-03-10 | 2018-10-02 | The Regents Of The University Of California | Methods of generating nucleic acid fragments |
| MX2012013037A (es) | 2010-05-10 | 2013-07-29 | Univ California | Composiciones de endorribonucleasa y metodos de uso de las mismas. |
| EP3156062A1 (en) | 2010-05-17 | 2017-04-19 | Sangamo BioSciences, Inc. | Novel dna-binding proteins and uses thereof |
| US20140148361A1 (en) | 2010-06-07 | 2014-05-29 | Barry L. Stoddard | Generation and Expression of Engineered I-ONUI Endonuclease and Its Homologues and Uses Thereof |
| CN103168101A (zh) | 2010-07-23 | 2013-06-19 | 西格马-奥尔德里奇有限责任公司 | 使用靶向核酸内切酶和单链核酸的基因组编辑 |
| DK2601611T3 (da) | 2010-08-02 | 2021-02-01 | Integrated Dna Tech Inc | Fremgangsmåder til forudsigelse af stabilitet og smeltetemperaturer for nukleinsyreduplekser |
| WO2012054726A1 (en) | 2010-10-20 | 2012-04-26 | Danisco A/S | Lactococcus crispr-cas sequences |
| KR20120096395A (ko) | 2011-02-22 | 2012-08-30 | 주식회사 툴젠 | 뉴클레아제에 의해 유전자 변형된 세포를 농축시키는 방법 |
| WO2012164565A1 (en) | 2011-06-01 | 2012-12-06 | Yeda Research And Development Co. Ltd. | Compositions and methods for downregulating prokaryotic genes |
| US9394545B2 (en) | 2011-09-21 | 2016-07-19 | Sangamo Biosciences, Inc. | Methods and compositions for regulation of transgene expression |
| US8450107B1 (en) | 2011-11-30 | 2013-05-28 | The Broad Institute Inc. | Nucleotide-specific recognition sequences for designer TAL effectors |
| US9046593B2 (en) | 2011-12-15 | 2015-06-02 | The Boeing Company | Method and apparatus for detecting and classifying signals |
| GB201122458D0 (en) | 2011-12-30 | 2012-02-08 | Univ Wageningen | Modified cascade ribonucleoproteins and uses thereof |
| WO2013126794A1 (en) | 2012-02-24 | 2013-08-29 | Fred Hutchinson Cancer Research Center | Compositions and methods for the treatment of hemoglobinopathies |
| NZ629427A (en) | 2012-02-29 | 2016-04-29 | Sangamo Biosciences Inc | Methods and compositions for treating huntington’s disease |
| WO2013141680A1 (en) | 2012-03-20 | 2013-09-26 | Vilnius University | RNA-DIRECTED DNA CLEAVAGE BY THE Cas9-crRNA COMPLEX |
| US9637739B2 (en) | 2012-03-20 | 2017-05-02 | Vilnius University | RNA-directed DNA cleavage by the Cas9-crRNA complex |
| AU2013204327B2 (en) | 2012-04-20 | 2016-09-01 | Aviagen | Cell transfection method |
| EP2841581B2 (en) | 2012-04-23 | 2023-03-08 | BASF Agricultural Solutions Seed US LLC | Targeted genome engineering in plants |
| EP2847335B1 (en) | 2012-04-25 | 2018-06-27 | Regeneron Pharmaceuticals, Inc. | Nuclease-mediated targeting with large targeting vectors |
| WO2013166315A1 (en) | 2012-05-02 | 2013-11-07 | Dow Agrosciences Llc | Targeted modification of malate dehydrogenase |
| KR102116153B1 (ko) | 2012-05-07 | 2020-05-27 | 상가모 테라퓨틱스, 인코포레이티드 | 도입유전자의 뉴클레아제-매개 표적화된 통합을 위한 방법 및 조성물 |
| US11120889B2 (en) | 2012-05-09 | 2021-09-14 | Georgia Tech Research Corporation | Method for synthesizing a nuclease with reduced off-site cleavage |
| LT2800811T (lt) * | 2012-05-25 | 2017-09-11 | The Regents Of The University Of California | Būdai ir kompozicijos, skirti tikslinės dnr modifikavimui, panaudojant adresuotą rnr, ir transkripcijos moduliavimui, panaudojant adresuotą rnr |
| WO2013181440A1 (en) | 2012-05-30 | 2013-12-05 | Baylor College Of Medicine | Supercoiled minivectors as a tool for dna repair, alteration and replacement |
| US9102936B2 (en) | 2012-06-11 | 2015-08-11 | Agilent Technologies, Inc. | Method of adaptor-dimer subtraction using a CRISPR CAS6 protein |
| WO2013188522A2 (en) | 2012-06-12 | 2013-12-19 | Genentech, Inc. | Methods and compositions for generating conditional knock-out alleles |
| EP2674501A1 (en) | 2012-06-14 | 2013-12-18 | Agence nationale de sécurité sanitaire de l'alimentation,de l'environnement et du travail | Method for detecting and identifying enterohemorrhagic Escherichia coli |
| WO2013188638A2 (en) | 2012-06-15 | 2013-12-19 | The Regents Of The University Of California | Endoribonucleases and methods of use thereof |
| WO2013192278A1 (en) | 2012-06-19 | 2013-12-27 | Regents Of The University Of Minnesota | Gene targeting in plants using dna viruses |
| EP2872154B1 (en) | 2012-07-11 | 2017-05-31 | Sangamo BioSciences, Inc. | Methods and compositions for delivery of biologics |
| WO2014011237A1 (en) | 2012-07-11 | 2014-01-16 | Sangamo Biosciences, Inc. | Methods and compositions for the treatment of lysosomal storage diseases |
| CN116622704A (zh) | 2012-07-25 | 2023-08-22 | 布罗德研究所有限公司 | 可诱导的dna结合蛋白和基因组干扰工具及其应用 |
| WO2014022702A2 (en) | 2012-08-03 | 2014-02-06 | The Regents Of The University Of California | Methods and compositions for controlling gene expression by rna processing |
| UA118957C2 (uk) | 2012-08-29 | 2019-04-10 | Сангамо Біосайєнсиз, Інк. | Спосіб і композиція для лікування генетичного захворювання |
| UA119135C2 (uk) | 2012-09-07 | 2019-05-10 | ДАУ АГРОСАЙЄНСІЗ ЕлЕлСі | Спосіб отримання трансгенної рослини |
| AU2013312538B2 (en) | 2012-09-07 | 2019-01-24 | Corteva Agriscience Llc | FAD3 performance loci and corresponding target site specific binding proteins capable of inducing targeted breaks |
| UA118090C2 (uk) | 2012-09-07 | 2018-11-26 | ДАУ АГРОСАЙЄНСІЗ ЕлЕлСі | Спосіб інтегрування послідовності нуклеїнової кислоти, що представляє інтерес, у ген fad2 у клітині сої та специфічний для локусу fad2 білок, що зв'язується, здатний індукувати спрямований розрив |
| EP3483185B1 (en) | 2012-10-12 | 2020-09-09 | The General Hospital Corporation | Transcription activator-like effector (tale) - lysine-specific demethylase 1 (lsd1) fusion proteins |
| AU2013335451C1 (en) | 2012-10-23 | 2024-07-04 | Toolgen Incorporated | Composition for cleaving a target DNA comprising a guide RNA specific for the target DNA and Cas protein-encoding nucleic acid or Cas protein, and use thereof |
| WO2014070887A1 (en) | 2012-10-30 | 2014-05-08 | Recombinetics, Inc. | Control of sexual maturation in animals |
| WO2014071006A1 (en) | 2012-10-31 | 2014-05-08 | Cellectis | Coupling herbicide resistance with targeted insertion of transgenes in plants |
| US20140127752A1 (en) | 2012-11-07 | 2014-05-08 | Zhaohui Zhou | Method, composition, and reagent kit for targeted genomic enrichment |
| EP3138911B1 (en) | 2012-12-06 | 2018-12-05 | Sigma Aldrich Co. LLC | Crispr-based genome modification and regulation |
| WO2014093479A1 (en) | 2012-12-11 | 2014-06-19 | Montana State University | Crispr (clustered regularly interspaced short palindromic repeats) rna-guided control of gene regulation |
| DK2931898T3 (en) | 2012-12-12 | 2016-06-20 | Massachusetts Inst Technology | CONSTRUCTION AND OPTIMIZATION OF SYSTEMS, PROCEDURES AND COMPOSITIONS FOR SEQUENCE MANIPULATION WITH FUNCTIONAL DOMAINS |
| WO2014093701A1 (en) | 2012-12-12 | 2014-06-19 | The Broad Institute, Inc. | Functional genomics using crispr-cas systems, compositions, methods, knock out libraries and applications thereof |
| CN110982844B (zh) | 2012-12-12 | 2024-08-13 | 布罗德研究所有限公司 | 用于序列操纵的crispr-cas组分系统、方法以及组合物 |
| US8697359B1 (en) | 2012-12-12 | 2014-04-15 | The Broad Institute, Inc. | CRISPR-Cas systems and methods for altering expression of gene products |
| EP2931892B1 (en) | 2012-12-12 | 2018-09-12 | The Broad Institute, Inc. | Methods, models, systems, and apparatus for identifying target sequences for cas enzymes or crispr-cas systems for target sequences and conveying results thereof |
| US20140179770A1 (en) | 2012-12-12 | 2014-06-26 | Massachusetts Institute Of Technology | Delivery, engineering and optimization of systems, methods and compositions for sequence manipulation and therapeutic applications |
| US20140310830A1 (en) | 2012-12-12 | 2014-10-16 | Feng Zhang | CRISPR-Cas Nickase Systems, Methods And Compositions For Sequence Manipulation in Eukaryotes |
| EP3705490B1 (en) | 2012-12-12 | 2024-03-06 | The Broad Institute, Inc. | Engineering and optimization of improved systems, methods and enzyme compositions for sequence manipulation |
| ES2576126T3 (es) | 2012-12-12 | 2016-07-05 | The Broad Institute, Inc. | Modificación por tecnología genética y optimización de sistemas, métodos y composiciones enzimáticas mejorados para la manipulación de secuencias |
| CN119752887A (zh) | 2012-12-12 | 2025-04-04 | 布罗德研究所有限公司 | 用于序列操纵的系统、方法和优化的指导组合物的工程化 |
| US9493844B2 (en) | 2012-12-13 | 2016-11-15 | Dow Agrosciences Llc | DNA detection methods for site specific nuclease activity |
| US8939652B2 (en) | 2012-12-13 | 2015-01-27 | Us Synthetic Corporation | Roller bearing apparatuses including compliant rolling elements, and related methods of manufacture |
| AR093946A1 (es) | 2012-12-13 | 2015-07-01 | Dow Agrosciences Llc | Un proceso mejorado para el aislamiento del acido 4-amino-3-cloro-6-(4-cloro-2-fluor-3-metoxi-fenil)piridina-2-carboxilico |
| SG10201704932UA (en) | 2012-12-17 | 2017-07-28 | Harvard College | Rna-guided human genome engineering |
| NL2010038C2 (en) | 2012-12-21 | 2014-06-24 | Koni Bv | Shock absorber. |
| ES2953523T3 (es) | 2012-12-27 | 2023-11-14 | Keygene Nv | Método para inducir una translocación dirigida en una planta |
| WO2014113493A1 (en) | 2013-01-16 | 2014-07-24 | Emory University | Cas9-nucleic acid complexes and uses related thereto |
| CN103233028B (zh) | 2013-01-25 | 2015-05-13 | 南京徇齐生物技术有限公司 | 一种无物种限制无生物安全性问题的真核生物基因打靶方法及螺旋结构dna序列 |
| WO2014127287A1 (en) | 2013-02-14 | 2014-08-21 | Massachusetts Institute Of Technology | Method for in vivo tergated mutagenesis |
| CA2900992C (en) | 2013-02-20 | 2023-02-28 | Regeneron Pharmaceuticals, Inc. | Genetic modification of rats |
| WO2014130955A1 (en) | 2013-02-25 | 2014-08-28 | Sangamo Biosciences, Inc. | Methods and compositions for enhancing nuclease-mediated gene disruption |
| US10760064B2 (en) | 2013-03-15 | 2020-09-01 | The General Hospital Corporation | RNA-guided targeting of genetic and epigenomic regulatory proteins to specific genomic loci |
| US20140273230A1 (en) | 2013-03-15 | 2014-09-18 | Sigma-Aldrich Co., Llc | Crispr-based genome modification and regulation |
| US9885033B2 (en) | 2013-03-15 | 2018-02-06 | The General Hospital Corporation | Increasing specificity for RNA-guided genome editing |
| US11332719B2 (en) | 2013-03-15 | 2022-05-17 | The Broad Institute, Inc. | Recombinant virus and preparations thereof |
| US9234213B2 (en) | 2013-03-15 | 2016-01-12 | System Biosciences, Llc | Compositions and methods directed to CRISPR/Cas genomic engineering systems |
| CA2906747A1 (en) | 2013-03-15 | 2014-09-18 | Regents Of The University Of Minnesota | Engineering plant genomes using crispr/cas systems |
| US20140364333A1 (en) | 2013-03-15 | 2014-12-11 | President And Fellows Of Harvard College | Methods for Live Imaging of Cells |
| JP2016522679A (ja) | 2013-04-04 | 2016-08-04 | プレジデント アンド フェローズ オブ ハーバード カレッジ | CRISPR/Cas系を用いたゲノム編集の治療的使用 |
| UA121197C2 (uk) | 2013-04-05 | 2020-04-27 | Доу Агросайенсіс Ллс | Нуклеаза "цинкові пальці" для модифікацїї гена ahas та спосіб її використання |
| PL3456831T3 (pl) | 2013-04-16 | 2021-12-06 | Regeneron Pharmaceuticals, Inc. | Ukierunkowana modyfikacja genomu szczura |
| CN103224947B (zh) | 2013-04-28 | 2015-06-10 | 陕西师范大学 | 一种基因打靶系统 |
| JP2016518142A (ja) | 2013-05-10 | 2016-06-23 | サンガモ バイオサイエンシーズ, インコーポレイテッド | ヌクレアーゼ媒介ゲノム遺伝子操作のための送達方法および組成物 |
| CA2913234A1 (en) | 2013-05-22 | 2014-11-27 | Northwestern University | Rna-directed dna cleavage and gene editing by cas9 enzyme from neisseria meningitidis |
| US10117124B2 (en) | 2013-05-28 | 2018-10-30 | Telefonaktiebolaget L M Ericsson (Publ) | Method, apparatus and computer program for updating a prioritization level of a service data flow based on traffic size per time unit of said data flow |
| US9873907B2 (en) | 2013-05-29 | 2018-01-23 | Agilent Technologies, Inc. | Method for fragmenting genomic DNA using CAS9 |
| US9267135B2 (en) | 2013-06-04 | 2016-02-23 | President And Fellows Of Harvard College | RNA-guided transcriptional regulation |
| DK3011032T3 (da) | 2013-06-17 | 2020-01-20 | Broad Inst Inc | Fremføring, modificering og optimering af systemer, fremgangsmåder og sammensætninger til målretning mod og modellering af sygdomme og forstyrrelser i postmitotiske celler |
| NL2010994C2 (en) | 2013-06-17 | 2014-12-18 | Fuji Seal Europe Bv | Container sleeving method and device. |
| WO2014204727A1 (en) | 2013-06-17 | 2014-12-24 | The Broad Institute Inc. | Functional genomics using crispr-cas systems, compositions methods, screens and applications thereof |
| KR20160030187A (ko) | 2013-06-17 | 2016-03-16 | 더 브로드 인스티튜트, 인코퍼레이티드 | 간의 표적화 및 치료를 위한 CRISPRCas 시스템, 벡터 및 조성물의 전달 및 용도 |
| EP3011030B1 (en) | 2013-06-17 | 2023-11-08 | The Broad Institute, Inc. | Optimized crispr-cas double nickase systems, methods and compositions for sequence manipulation |
| EP3011035B1 (en) | 2013-06-17 | 2020-05-13 | The Broad Institute, Inc. | Assay for quantitative evaluation of target site cleavage by one or more crispr-cas guide sequences |
| BR122021009076B1 (pt) | 2013-06-17 | 2024-02-15 | The Broad Institute Inc. | Vetor viral contendo molécula(s) de ácido nucleico heterólogo, composição, uso e métodos do mesmo |
| KR20160044457A (ko) | 2013-06-17 | 2016-04-25 | 더 브로드 인스티튜트, 인코퍼레이티드 | 서열 조작을 위한 탠덤 안내 시스템, 방법 및 조성물의 전달, 조작 및 최적화 |
| CN103343120B (zh) | 2013-07-04 | 2015-03-04 | 中国科学院遗传与发育生物学研究所 | 一种小麦基因组定点改造方法 |
| CA3218040A1 (en) | 2013-07-10 | 2015-01-15 | President And Fellows Of Harvard College | Orthogonal cas9 proteins for rna-guided gene regulation and editing |
| US10421957B2 (en) | 2013-07-29 | 2019-09-24 | Agilent Technologies, Inc. | DNA assembly using an RNA-programmable nickase |
| UY35812A (es) | 2013-11-04 | 2015-05-29 | Dow Agrosciences Llc | ?loci de maíz óptimos?. |
| WO2015066643A1 (en) | 2013-11-04 | 2015-05-07 | Dow Agrosciences Llc | Optimal soybean loci |
| CN105980395A (zh) | 2013-11-04 | 2016-09-28 | 美国陶氏益农公司 | 最优大豆座位 |
| US9834791B2 (en) | 2013-11-07 | 2017-12-05 | Editas Medicine, Inc. | CRISPR-related methods and compositions with governing gRNAS |
| JP2016537028A (ja) | 2013-11-18 | 2016-12-01 | クリスパー セラピューティクス アーゲー | Crispr−casシステムの材料及び方法 |
| US10787684B2 (en) | 2013-11-19 | 2020-09-29 | President And Fellows Of Harvard College | Large gene excision and insertion |
| MX388127B (es) | 2013-12-11 | 2025-03-19 | Regeneron Pharma | Metodos y composiciones para la modificacion dirigida de un genoma. |
| EP3079726B1 (en) | 2013-12-12 | 2018-12-05 | The Broad Institute, Inc. | Delivery, use and therapeutic applications of the crispr-cas systems and compositions for targeting disorders and diseases using particle delivery components |
| US20150166985A1 (en) | 2013-12-12 | 2015-06-18 | President And Fellows Of Harvard College | Methods for correcting von willebrand factor point mutations |
| AU2014370416B2 (en) | 2013-12-26 | 2021-03-11 | The General Hospital Corporation | Multiplex guide RNAs |
| WO2015116686A1 (en) | 2014-01-29 | 2015-08-06 | Agilent Technologies, Inc. | Cas9-based isothermal method of detection of specific dna sequence |
| US20150291969A1 (en) | 2014-01-30 | 2015-10-15 | Chromatin, Inc. | Compositions for reduced lignin content in sorghum and improving cell wall digestibility, and methods of making the same |
| US20150225801A1 (en) | 2014-02-11 | 2015-08-13 | California Institute Of Technology | Recording and mapping lineage information and molecular events in individual cells |
| WO2015127439A1 (en) | 2014-02-24 | 2015-08-27 | Sangamo Biosciences, Inc. | Methods and compositions for nuclease-mediated targeted integration |
| CA2942762C (en) | 2014-03-18 | 2023-10-17 | Sangamo Biosciences, Inc. | Methods and compositions for regulation of zinc finger protein expression |
| EP3498845B1 (en) | 2014-04-01 | 2022-06-22 | Editas Medicine, Inc. | Crispr/cas-related methods and compositions for treating herpes simplex virus type 1 (hsv-1) |
| DE102015226614A1 (de) | 2015-12-23 | 2017-06-29 | Robert Bosch Gmbh | Verfahren zum Betreiben eines Kraftfahrzeugs, Steuerungseinheit für ein Antriebssystem und ein Antriebssystem |
| US10250380B2 (en) | 2016-12-12 | 2019-04-02 | Qualcomm Incorporated | Techniques for unified synchronization channel design in new radio |
| US11352698B2 (en) | 2019-04-25 | 2022-06-07 | Samsung Electronics Co., Ltd. | Atomic layer deposition apparatus and methods of fabricating semiconductor devices using the same |
-
2013
- 2013-03-15 LT LTEP13793997.1T patent/LT2800811T/lt unknown
- 2013-03-15 ES ES17163434.8T patent/ES2670718T3/es active Active
- 2013-03-15 PT PT137939971T patent/PT2800811T/pt unknown
- 2013-03-15 EP EP25159662.3A patent/EP4570908A2/en active Pending
- 2013-03-15 FI FIEP23187511.3T patent/FI4289948T3/fi active
- 2013-03-15 LT LTEP18152360.6T patent/LT3401400T/lt unknown
- 2013-03-15 EP EP21207127.8A patent/EP4043564A1/en not_active Withdrawn
- 2013-03-15 HU HUE18152360A patent/HUE043861T2/hu unknown
- 2013-03-15 PE PE2019000943A patent/PE20190842A1/es unknown
- 2013-03-15 HR HRP20250549TT patent/HRP20250549T1/hr unknown
- 2013-03-15 ES ES13793997.1T patent/ES2636902T3/es active Active
- 2013-03-15 SG SG10201809817UA patent/SG10201809817UA/en unknown
- 2013-03-15 PL PL23187511.3T patent/PL4289948T3/pl unknown
- 2013-03-15 HU HUE17163434A patent/HUE038850T2/hu unknown
- 2013-03-15 ME MEP-2019-155A patent/ME03530B/me unknown
- 2013-03-15 WO PCT/US2013/032589 patent/WO2013176772A1/en active Application Filing
- 2013-03-15 DE DE202013012240.4U patent/DE202013012240U1/de not_active Expired - Lifetime
- 2013-03-15 KR KR1020177034069A patent/KR20170134766A/ko not_active Ceased
- 2013-03-15 MY MYPI2014003102A patent/MY184753A/en unknown
- 2013-03-15 EP EP18152360.6A patent/EP3401400B1/en not_active Revoked
- 2013-03-15 US US13/842,859 patent/US10266850B2/en active Active
- 2013-03-15 RS RS20170783A patent/RS56119B1/sr unknown
- 2013-03-15 DK DK18152360.6T patent/DK3401400T3/da active
- 2013-03-15 LT LTEP23187511.3T patent/LT4289948T/lt unknown
- 2013-03-15 ME MEP-2017-180A patent/ME02836B/me unknown
- 2013-03-15 JP JP2015514015A patent/JP6343605B2/ja active Active
- 2013-03-15 FI FIEP19157590.1T patent/FI3597749T3/fi active
- 2013-03-15 KR KR1020147036096A patent/KR20150016588A/ko not_active Ceased
- 2013-03-15 PT PT171634348T patent/PT3241902T/pt unknown
- 2013-03-15 MX MX2017010309A patent/MX362866B/es unknown
- 2013-03-15 EP EP19157590.1A patent/EP3597749B1/en active Active
- 2013-03-15 NZ NZ728024A patent/NZ728024A/en unknown
- 2013-03-15 ES ES19157590T patent/ES2960803T3/es active Active
- 2013-03-15 CA CA2872241A patent/CA2872241C/en active Active
- 2013-03-15 BR BR112014029441-0A patent/BR112014029441B1/pt active IP Right Grant
- 2013-03-15 GB GB1420270.9A patent/GB2518764C/en active Active
- 2013-03-15 LT LTEP19157590.1T patent/LT3597749T/lt unknown
- 2013-03-15 MY MYPI2018700285A patent/MY189533A/en unknown
- 2013-03-15 DK DK17163434.8T patent/DK3241902T3/en active
- 2013-03-15 CN CN201380038920.6A patent/CN104854241B/zh active Active
- 2013-03-15 PT PT191575901T patent/PT3597749T/pt unknown
- 2013-03-15 HK HK15104473.1A patent/HK1204003A1/xx unknown
- 2013-03-15 DE DE202013012242.0U patent/DE202013012242U1/de not_active Expired - Lifetime
- 2013-03-15 MX MX2014014477A patent/MX349744B/es active IP Right Grant
- 2013-03-15 PE PE2019000944A patent/PE20190843A1/es unknown
- 2013-03-15 PL PL13793997T patent/PL2800811T3/pl unknown
- 2013-03-15 DK DK23187511.3T patent/DK4289948T3/da active
- 2013-03-15 RS RSP20190673 patent/RS59199B1/sr unknown
- 2013-03-15 RS RS20250443A patent/RS66774B1/sr unknown
- 2013-03-15 SI SI201331048T patent/SI3241902T1/en unknown
- 2013-03-15 GB GB1601071.2A patent/GB2537000C/en active Active
- 2013-03-15 SG SG11201407702XA patent/SG11201407702XA/en unknown
- 2013-03-15 EP EP13793997.1A patent/EP2800811B1/en not_active Revoked
- 2013-03-15 DK DK13793997.1T patent/DK2800811T3/en active
- 2013-03-15 TR TR2018/06812T patent/TR201806812T4/tr unknown
- 2013-03-15 NZ NZ714353A patent/NZ714353A/en unknown
- 2013-03-15 HU HUE19157590A patent/HUE064300T2/hu unknown
- 2013-03-15 SI SI201331467T patent/SI3401400T1/sl unknown
- 2013-03-15 SI SI201332059T patent/SI3597749T1/sl unknown
- 2013-03-15 PE PE2014002211A patent/PE20150336A1/es active IP Right Grant
- 2013-03-15 DE DE202013012241.2U patent/DE202013012241U1/de not_active Expired - Lifetime
- 2013-03-15 PL PL17163434T patent/PL3241902T3/pl unknown
- 2013-03-15 RS RS20180482A patent/RS57287B1/sr unknown
- 2013-03-15 PL PL18152360T patent/PL3401400T3/pl unknown
- 2013-03-15 ES ES18152360T patent/ES2728782T3/es active Active
- 2013-03-15 SG SG10201701800YA patent/SG10201701800YA/en unknown
- 2013-03-15 GE GEAP201313674A patent/GEP20217251B/en unknown
- 2013-03-15 PT PT231875113T patent/PT4289948T/pt unknown
- 2013-03-15 EP EP17163434.8A patent/EP3241902B1/en not_active Revoked
- 2013-03-15 RS RS20230851A patent/RS64622B1/sr unknown
- 2013-03-15 MA MA37663A patent/MA37663B1/fr unknown
- 2013-03-15 SI SI201330747T patent/SI2800811T1/sl unknown
- 2013-03-15 PL PL19157590.1T patent/PL3597749T3/pl unknown
- 2013-03-15 HR HRP20171163TT patent/HRP20171163T1/hr unknown
- 2013-03-15 EP EP23187511.3A patent/EP4289948B1/en active Active
- 2013-03-15 AU AU2013266968A patent/AU2013266968B2/en active Active
- 2013-03-15 US US14/403,475 patent/US20160046961A1/en not_active Abandoned
- 2013-03-15 EA EA201401319A patent/EA038924B1/ru unknown
- 2013-03-15 DK DK19157590.1T patent/DK3597749T5/da active
- 2013-03-15 CN CN201710585690.5A patent/CN107603976B/zh active Active
- 2013-03-15 HR HRP20231042TT patent/HRP20231042T1/hr unknown
- 2013-03-15 LT LTEP17163434.8T patent/LT3241902T/lt unknown
- 2013-03-15 UA UAA201413835A patent/UA118014C2/uk unknown
- 2013-03-15 ES ES23187511T patent/ES3028090T3/es active Active
- 2013-03-15 PT PT18152360T patent/PT3401400T/pt unknown
- 2013-03-15 PE PE2019000945A patent/PE20190844A1/es unknown
- 2013-03-15 MX MX2019001995A patent/MX369077B/es unknown
-
2014
- 2014-11-02 IL IL235461A patent/IL235461B/en active IP Right Grant
- 2014-11-19 PH PH12014502574A patent/PH12014502574B1/en unknown
- 2014-11-24 CR CR20140538A patent/CR20140538A/es unknown
- 2014-11-25 MX MX2019012772A patent/MX2019012772A/es unknown
- 2014-11-25 CL CL2014003208A patent/CL2014003208A1/es unknown
- 2014-11-25 EC ECIEPI201428704A patent/ECSP14028704A/es unknown
- 2014-11-25 CO CO14259531A patent/CO7151523A2/es unknown
- 2014-11-25 TN TN2014000493A patent/TN2014000493A1/fr unknown
-
2015
- 2015-04-13 US US14/685,513 patent/US20160060653A1/en not_active Abandoned
- 2015-04-13 US US14/685,504 patent/US10301651B2/en active Active
- 2015-04-13 US US14/685,502 patent/US10000772B2/en active Active
- 2015-04-13 US US14/685,514 patent/US20160060654A1/en not_active Abandoned
- 2015-04-13 US US14/685,516 patent/US20160068864A1/en not_active Abandoned
- 2015-07-03 GB GBGB1511669.2A patent/GB201511669D0/en not_active Ceased
- 2015-11-16 US US14/942,782 patent/US10227611B2/en active Active
-
2016
- 2016-04-04 US US15/090,511 patent/US20170051312A1/en not_active Abandoned
- 2016-04-26 US US15/138,604 patent/US10113167B2/en active Active
-
2017
- 2017-02-16 US US15/435,233 patent/US10407697B2/en active Active
- 2017-08-08 CY CY20171100846T patent/CY1119282T1/el unknown
- 2017-09-07 AU AU2017225060A patent/AU2017225060B2/en active Active
- 2017-11-03 US US15/803,424 patent/US10519467B2/en active Active
-
2018
- 2018-03-07 US US15/915,020 patent/US20190062790A1/en not_active Abandoned
- 2018-03-19 US US15/925,544 patent/US10385360B2/en active Active
- 2018-04-06 US US15/947,680 patent/US20180230495A1/en active Pending
- 2018-04-06 US US15/947,718 patent/US20180230497A1/en active Pending
- 2018-04-06 US US15/947,700 patent/US20180230496A1/en active Pending
- 2018-04-23 US US15/959,735 patent/US20180298407A1/en not_active Abandoned
- 2018-04-23 US US15/959,762 patent/US20180237801A1/en not_active Abandoned
- 2018-04-23 US US15/959,715 patent/US20180298406A1/en not_active Abandoned
- 2018-04-23 US US15/959,782 patent/US20180245100A1/en not_active Abandoned
- 2018-04-23 US US15/959,802 patent/US20180273981A1/en not_active Abandoned
- 2018-04-27 US US15/965,598 patent/US20180245101A1/en not_active Abandoned
- 2018-04-27 US US15/965,603 patent/US20180251791A1/en not_active Abandoned
- 2018-05-16 US US15/981,808 patent/US20180251794A1/en active Pending
- 2018-05-16 US US15/981,809 patent/US20180251795A1/en active Pending
- 2018-05-16 US US15/981,807 patent/US20180251793A1/en active Pending
- 2018-05-16 CY CY20181100506T patent/CY1120291T1/el unknown
- 2018-05-21 JP JP2018097369A patent/JP6692856B2/ja active Active
- 2018-05-21 HR HRP20180794TT patent/HRP20180794T1/hr unknown
- 2018-07-11 US US16/033,016 patent/US10308961B2/en active Active
- 2018-07-11 US US16/033,005 patent/US10415061B2/en active Active
- 2018-07-11 US US16/033,002 patent/US10421980B2/en active Active
- 2018-09-03 IL IL261563A patent/IL261563A/en unknown
- 2018-09-03 IL IL261566A patent/IL261566B/en unknown
- 2018-09-03 IL IL261568A patent/IL261568B/en unknown
- 2018-09-03 IL IL261570A patent/IL261570B/en unknown
- 2018-09-03 IL IL261567A patent/IL261567B/en active IP Right Grant
- 2018-09-03 IL IL261569A patent/IL261569B/en unknown
- 2018-09-03 IL IL261565A patent/IL261565B/en unknown
- 2018-09-19 US US16/136,168 patent/US20190002923A1/en active Pending
- 2018-09-19 US US16/136,165 patent/US20190002922A1/en active Pending
- 2018-09-19 US US16/136,159 patent/US20190002921A1/en active Pending
- 2018-09-19 US US16/136,175 patent/US20190010520A1/en active Pending
- 2018-11-27 US US16/201,865 patent/US10669560B2/en active Active
- 2018-11-27 US US16/201,855 patent/US10351878B2/en active Active
- 2018-11-27 US US16/201,853 patent/US10358659B2/en active Active
- 2018-11-27 US US16/201,848 patent/US10337029B2/en active Active
- 2018-11-27 US US16/201,862 patent/US10626419B2/en active Active
- 2018-11-27 US US16/201,836 patent/US10358658B2/en active Active
-
2019
- 2019-02-14 US US16/276,343 patent/US10400253B2/en active Active
- 2019-02-14 US US16/276,348 patent/US10428352B2/en active Active
- 2019-02-14 US US16/276,361 patent/US20190169645A1/en active Pending
- 2019-02-14 US US16/276,356 patent/US10443076B2/en active Active
- 2019-02-14 US US16/276,365 patent/US20190169646A1/en active Pending
- 2019-02-14 US US16/276,374 patent/US20190169648A1/en active Pending
- 2019-02-14 US US16/276,368 patent/US20190169647A1/en active Pending
- 2019-02-14 US US16/276,352 patent/US10640791B2/en active Active
- 2019-02-15 US US16/277,090 patent/US10487341B2/en active Active
- 2019-03-18 AU AU2019201850A patent/AU2019201850B2/en active Active
- 2019-04-10 US US16/380,781 patent/US10533190B2/en active Active
- 2019-04-10 US US16/380,758 patent/US10526619B2/en active Active
- 2019-04-11 US US16/382,097 patent/US10563227B2/en active Active
- 2019-04-11 US US16/382,096 patent/US10550407B2/en active Active
- 2019-04-11 US US16/382,093 patent/US10513712B2/en active Active
- 2019-04-11 US US16/382,100 patent/US10570419B2/en active Active
- 2019-04-12 US US16/383,422 patent/US10577631B2/en active Active
- 2019-04-12 US US16/383,443 patent/US10597680B2/en active Active
- 2019-04-12 US US16/383,412 patent/US10612045B2/en active Active
- 2019-04-12 US US16/383,433 patent/US10676759B2/en active Active
- 2019-04-16 US US16/385,360 patent/US20200071728A1/en not_active Abandoned
- 2019-04-16 US US16/385,383 patent/US11634730B2/en active Active
- 2019-05-20 CY CY20191100536T patent/CY1121657T1/el unknown
- 2019-05-28 HR HRP20190965TT patent/HRP20190965T1/hr unknown
- 2019-11-21 JP JP2019210828A patent/JP6887479B2/ja active Active
-
2020
- 2020-02-13 US US16/790,368 patent/US10752920B2/en active Active
- 2020-03-20 US US16/825,807 patent/US20200277631A1/en not_active Abandoned
- 2020-04-27 US US16/859,182 patent/US20200308605A1/en not_active Abandoned
- 2020-06-04 US US16/892,663 patent/US10793878B1/en active Active
- 2020-06-04 US US16/892,631 patent/US10774344B1/en active Active
- 2020-06-10 US US16/898,197 patent/US20200308607A1/en not_active Abandoned
- 2020-06-10 US US16/898,161 patent/US20200325495A1/en not_active Abandoned
- 2020-06-10 US US16/898,186 patent/US20200308606A1/en not_active Abandoned
- 2020-07-21 US US16/935,011 patent/US10988780B2/en active Active
- 2020-07-21 US US16/935,016 patent/US10982230B2/en active Active
- 2020-07-21 US US16/935,023 patent/US10982231B2/en active Active
- 2020-07-21 US US16/935,007 patent/US10900054B2/en active Active
- 2020-10-23 US US17/079,070 patent/US11242543B2/en active Active
- 2020-10-29 US US17/084,023 patent/US11028412B2/en active Active
- 2020-10-29 US US17/084,014 patent/US11186849B2/en active Active
- 2020-10-29 US US17/084,020 patent/US10988782B2/en active Active
- 2020-11-23 US US17/102,050 patent/US11001863B2/en active Active
- 2020-11-23 US US17/102,059 patent/US11008590B2/en active Active
- 2020-11-23 US US17/102,031 patent/US11008589B2/en active Active
-
2021
- 2021-02-12 AU AU2021200952A patent/AU2021200952B2/en not_active Expired - Fee Related
- 2021-04-01 US US17/220,693 patent/US11814645B2/en active Active
- 2021-04-01 US US17/220,692 patent/US11674159B2/en active Active
- 2021-05-18 JP JP2021083800A patent/JP2021121205A/ja not_active Withdrawn
- 2021-05-21 US US17/326,791 patent/US11293034B2/en active Active
- 2021-05-21 US US17/326,818 patent/US11274318B2/en active Active
- 2021-05-21 US US17/326,805 patent/US11401532B2/en active Active
- 2021-06-15 US US17/348,596 patent/US12180503B2/en active Active
- 2021-06-15 US US17/348,590 patent/US20210310028A1/en not_active Abandoned
- 2021-06-15 US US17/348,619 patent/US12180504B2/en active Active
- 2021-09-21 US US17/481,052 patent/US11332761B2/en active Active
- 2021-09-21 US US17/481,085 patent/US12123015B2/en active Active
- 2021-09-21 US US17/481,063 patent/US11970711B2/en active Active
- 2021-10-29 US US17/514,919 patent/US20220119846A1/en active Pending
- 2021-10-29 US US17/514,929 patent/US20220119847A1/en active Pending
- 2021-10-29 US US17/514,940 patent/US20220112521A1/en active Pending
- 2021-10-29 US US17/514,883 patent/US20220119845A1/en not_active Abandoned
- 2021-10-29 US US17/514,936 patent/US20220119848A1/en active Pending
- 2021-10-29 US US17/514,908 patent/US20220127645A1/en not_active Abandoned
- 2021-10-29 US US17/514,893 patent/US20220073952A1/en not_active Abandoned
- 2021-12-22 US US17/559,860 patent/US12215343B2/en active Active
-
2022
- 2022-03-24 US US17/703,845 patent/US11473108B2/en active Active
- 2022-03-24 US US17/703,861 patent/US11479794B2/en active Active
- 2022-03-24 US US17/703,849 patent/US11549127B2/en active Active
- 2022-11-10 US US18/054,538 patent/US20230227859A1/en not_active Abandoned
- 2022-12-09 US US18/064,011 patent/US20230212614A1/en not_active Abandoned
-
2023
- 2023-06-30 JP JP2023107774A patent/JP2023123755A/ja active Pending
- 2023-10-11 AU AU2023248113A patent/AU2023248113A1/en active Pending
-
2024
- 2024-06-24 US US18/751,730 patent/US20240417756A1/en active Pending
- 2024-10-25 US US18/926,911 patent/US20250122537A1/en active Pending
-
2025
- 2025-01-03 US US19/009,690 patent/US20250146024A1/en active Pending
Cited By (25)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| KR20150056539A (ko) * | 2012-07-25 | 2015-05-26 | 더 브로드 인스티튜트, 인코퍼레이티드 | 유도 dna 결합 단백질 및 게놈 교란 도구 및 이의 적용 |
| KR20220057633A (ko) * | 2012-10-23 | 2022-05-09 | 주식회사 툴젠 | 표적 DNA에 특이적인 가이드 RNA 및 Cas 단백질을 암호화하는 핵산 또는 Cas 단백질을 포함하는, 표적 DNA를 절단하기 위한 조성물 및 이의 용도 |
| KR20150101476A (ko) * | 2012-10-23 | 2015-09-03 | 주식회사 툴젠 | 표적 DNA에 특이적인 가이드 RNA 및 Cas 단백질을 암호화하는 핵산 또는 Cas 단백질을 포함하는, 표적 DNA를 절단하기 위한 조성물 및 이의 용도 |
| KR20150101477A (ko) * | 2012-10-23 | 2015-09-03 | 주식회사 툴젠 | 표적 DNA에 특이적인 가이드 RNA 및 Cas 단백질을 암호화하는 핵산 또는 Cas 단백질을 포함하는, 표적 DNA를 절단하기 위한 조성물 및 이의 용도 |
| KR20150101478A (ko) * | 2012-10-23 | 2015-09-03 | 주식회사 툴젠 | 표적 DNA에 특이적인 가이드 RNA 및 Cas 단백질을 암호화하는 핵산 또는 Cas 단백질을 포함하는, 표적 DNA를 절단하기 위한 조성물 및 이의 용도 |
| KR20230066138A (ko) * | 2012-10-23 | 2023-05-12 | 주식회사 툴젠 | 표적 DNA에 특이적인 가이드 RNA 및 Cas 단백질을 암호화하는 핵산 또는 Cas 단백질을 포함하는, 표적 DNA를 절단하기 위한 조성물 및 이의 용도 |
| KR20230064634A (ko) * | 2012-10-23 | 2023-05-10 | 주식회사 툴젠 | 표적 DNA에 특이적인 가이드 RNA 및 Cas 단백질을 암호화하는 핵산 또는 Cas 단백질을 포함하는, 표적 DNA를 절단하기 위한 조성물 및 이의 용도 |
| US10851380B2 (en) | 2012-10-23 | 2020-12-01 | Toolgen Incorporated | Methods for cleaving a target DNA using a guide RNA specific for the target DNA and Cas protein-encoding nucleic acid or Cas protein |
| KR20210013288A (ko) * | 2012-10-23 | 2021-02-03 | 주식회사 툴젠 | 표적 DNA에 특이적인 가이드 RNA 및 Cas 단백질을 암호화하는 핵산 또는 Cas 단백질을 포함하는, 표적 DNA를 절단하기 위한 조성물 및 이의 용도 |
| KR20170124565A (ko) * | 2015-03-03 | 2017-11-10 | 더 제너럴 하스피탈 코포레이션 | 변경된 PAM 특이성을 갖는 조작된 CRISPR-Cas9 뉴클레아제 |
| KR20180085795A (ko) * | 2015-12-04 | 2018-07-27 | 카리부 바이오사이언시스 인코포레이티드 | 조작된 핵산 표적화 핵산 |
| US12012598B2 (en) | 2016-08-12 | 2024-06-18 | Toolgen Incorporated | Manipulated immunoregulatory element and immunity altered thereby |
| WO2018208067A1 (ko) * | 2017-05-08 | 2018-11-15 | 주식회사 툴젠 | 인위적으로 조작된 조작면역세포 |
| US12275963B2 (en) | 2017-05-08 | 2025-04-15 | Toolgen Incorporated | Artificially manipulated immune cell |
| KR20200016892A (ko) * | 2017-06-09 | 2020-02-17 | 에디타스 메디신, 인코포레이티드 | 조작된 cas9 뉴클레아제 |
| US12297466B2 (en) | 2017-06-09 | 2025-05-13 | Editas Medicine, Inc. | Engineered Cas9 nucleases |
| KR20190046071A (ko) | 2017-10-25 | 2019-05-07 | 성균관대학교산학협력단 | 노화 모델 제조 방법 및 이에 의해 제조된 세포 또는 동물 노화 모델 |
| KR20200123255A (ko) * | 2018-03-26 | 2020-10-28 | 고쿠리츠다이가쿠호진 고베다이가쿠 | 세포에서 이중 가닥 dna의 표적 부위를 변형시키기 위한 방법 |
| KR20230022743A (ko) | 2021-08-09 | 2023-02-16 | 경상국립대학교산학협력단 | 콩 유전자교정 효율 증대를 위한 유전자교정 시스템 및 이의 용도 |
| KR20230022745A (ko) | 2021-08-09 | 2023-02-16 | 경상국립대학교산학협력단 | P34와 이의 상동체 동시 타겟 유전자교정 시스템 및 이의 용도 |
| KR20230022747A (ko) | 2021-08-09 | 2023-02-16 | 경상국립대학교산학협력단 | Mips1과 이의 상동체 동시 타겟 유전자교정 시스템 및 이의 용도 |
| KR20230022748A (ko) | 2021-08-09 | 2023-02-16 | 경상국립대학교산학협력단 | E2와 이의 상동체 동시 타겟 유전자교정 시스템 및 이의 용도 |
| KR20230022749A (ko) | 2021-08-09 | 2023-02-16 | 경상국립대학교산학협력단 | GmIPK1 유전자교정 시스템 및 이의 용도 |
| KR20230051095A (ko) | 2021-10-06 | 2023-04-17 | 주식회사 진코어 | 유전자 편집을 위한 TaRGET 시스템 및 이의 용도 |
| KR102703683B1 (ko) | 2021-10-06 | 2024-09-09 | 주식회사 진코어 | 유전자 편집을 위한 TaRGET 시스템 및 이의 용도 |
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| AU2021200952B2 (en) | Methods and compositions for RNA-directed target DNA modification and for RNA-directed modulation of transcription | |
| HK40071464A (en) | Methods and compositions for rna-directed target dna modification and for rna-directed modulation of transcription | |
| HK40012638A (en) | Methods and compositions for rna-directed target dna modification and for rna-directed modulation of transcription | |
| HK1260088A1 (en) | Methods and compositions for rna-directed target dna modification and for rna-directed modulation of transcription | |
| HK1260088B (en) | Methods and compositions for rna-directed target dna modification and for rna-directed modulation of transcription | |
| HK1230233A (en) | Methods and compositions for rna-directed target dna modification and for rna-directed modulation of transcription | |
| HK1230233A1 (en) | Methods and compositions for rna-directed target dna modification and for rna-directed modulation of transcription |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| PA0105 | International application |
Patent event date: 20141223 Patent event code: PA01051R01D Comment text: International Patent Application |
|
| PG1501 | Laying open of application | ||
| A201 | Request for examination | ||
| A302 | Request for accelerated examination | ||
| PA0201 | Request for examination |
Patent event code: PA02012R01D Patent event date: 20170307 Comment text: Request for Examination of Application |
|
| PA0302 | Request for accelerated examination |
Patent event date: 20170307 Patent event code: PA03022R01D Comment text: Request for Accelerated Examination |
|
| E902 | Notification of reason for refusal | ||
| PE0902 | Notice of grounds for rejection |
Comment text: Notification of reason for refusal Patent event date: 20170601 Patent event code: PE09021S01D |
|
| A107 | Divisional application of patent | ||
| PA0104 | Divisional application for international application |
Comment text: Divisional Application for International Patent Patent event code: PA01041R01D Patent event date: 20171124 |
|
| E601 | Decision to refuse application | ||
| PE0601 | Decision on rejection of patent |
Patent event date: 20171215 Comment text: Decision to Refuse Application Patent event code: PE06012S01D Patent event date: 20170601 Comment text: Notification of reason for refusal Patent event code: PE06011S01I |