JP2013507145A - 1,4−ブタンジオール、4−ヒドロキシブタナール、4−ヒドロキシブチリル−CoA、プトレシン及び関連化合物の生成のための微生物体並びに関連する方法 - Google Patents
1,4−ブタンジオール、4−ヒドロキシブタナール、4−ヒドロキシブチリル−CoA、プトレシン及び関連化合物の生成のための微生物体並びに関連する方法 Download PDFInfo
- Publication number
- JP2013507145A JP2013507145A JP2012534339A JP2012534339A JP2013507145A JP 2013507145 A JP2013507145 A JP 2013507145A JP 2012534339 A JP2012534339 A JP 2012534339A JP 2012534339 A JP2012534339 A JP 2012534339A JP 2013507145 A JP2013507145 A JP 2013507145A
- Authority
- JP
- Japan
- Prior art keywords
- microbial organism
- dehydrogenase
- putrescine
- pathway
- coa
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Pending
Links
- KIDHWZJUCRJVML-UHFFFAOYSA-N putrescine Chemical compound NCCCCN KIDHWZJUCRJVML-UHFFFAOYSA-N 0.000 title claims abstract description 365
- PIAOXUVIBAKVSP-UHFFFAOYSA-N γ-hydroxybutyraldehyde Chemical compound OCCCC=O PIAOXUVIBAKVSP-UHFFFAOYSA-N 0.000 title claims abstract description 186
- 239000005700 Putrescine Substances 0.000 title claims abstract description 179
- BAMBWCGEVIAQBF-CITAKDKDSA-N 4-hydroxybutyryl-CoA Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCSC(=O)CCCO)O[C@H]1N1C2=NC=NC(N)=C2N=C1 BAMBWCGEVIAQBF-CITAKDKDSA-N 0.000 title claims abstract description 146
- 238000004519 manufacturing process Methods 0.000 title claims abstract description 85
- 244000005700 microbiome Species 0.000 title claims abstract description 57
- 238000000034 method Methods 0.000 title claims description 63
- WERYXYBDKMZEQL-UHFFFAOYSA-N butane-1,4-diol Chemical compound OCCCCO WERYXYBDKMZEQL-UHFFFAOYSA-N 0.000 title abstract description 440
- 150000001875 compounds Chemical class 0.000 title description 34
- 230000037361 pathway Effects 0.000 claims abstract description 308
- 230000000813 microbial effect Effects 0.000 claims abstract description 299
- 102000004190 Enzymes Human genes 0.000 claims abstract description 237
- 108090000790 Enzymes Proteins 0.000 claims abstract description 237
- 150000007523 nucleic acids Chemical class 0.000 claims abstract description 197
- 102000039446 nucleic acids Human genes 0.000 claims abstract description 190
- 108020004707 nucleic acids Proteins 0.000 claims abstract description 190
- 102000004316 Oxidoreductases Human genes 0.000 claims description 191
- 108090000854 Oxidoreductases Proteins 0.000 claims description 191
- 101710088194 Dehydrogenase Proteins 0.000 claims description 180
- 230000015572 biosynthetic process Effects 0.000 claims description 97
- BTCSSZJGUNDROE-UHFFFAOYSA-N gamma-aminobutyric acid Chemical compound NCCCC(O)=O BTCSSZJGUNDROE-UHFFFAOYSA-N 0.000 claims description 64
- 108090000340 Transaminases Proteins 0.000 claims description 56
- MMTYWJNSFWVPPS-UHFFFAOYSA-N 5-hydroxy-2-oxopentanoic acid Chemical compound OCCCC(=O)C(O)=O MMTYWJNSFWVPPS-UHFFFAOYSA-N 0.000 claims description 48
- VNOYUJKHFWYWIR-ITIYDSSPSA-N succinyl-CoA Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCSC(=O)CCC(O)=O)O[C@H]1N1C2=NC=NC(N)=C2N=C1 VNOYUJKHFWYWIR-ITIYDSSPSA-N 0.000 claims description 44
- 108010021680 2-oxoglutarate decarboxylase Proteins 0.000 claims description 38
- SJZRECIVHVDYJC-UHFFFAOYSA-M 4-hydroxybutyrate Chemical compound OCCCC([O-])=O SJZRECIVHVDYJC-UHFFFAOYSA-M 0.000 claims description 37
- 108010093796 4-hydroxybutyrate dehydrogenase Proteins 0.000 claims description 32
- 238000012258 culturing Methods 0.000 claims description 31
- KPGXRSRHYNQIFN-UHFFFAOYSA-L 2-oxoglutarate(2-) Chemical compound [O-]C(=O)CCC(=O)C([O-])=O KPGXRSRHYNQIFN-UHFFFAOYSA-L 0.000 claims description 26
- 102000004031 Carboxy-Lyases Human genes 0.000 claims description 26
- 108090000489 Carboxy-Lyases Proteins 0.000 claims description 26
- WHUUTDBJXJRKMK-VKHMYHEASA-N L-glutamic acid Chemical compound OC(=O)[C@@H](N)CCC(O)=O WHUUTDBJXJRKMK-VKHMYHEASA-N 0.000 claims description 24
- 238000006114 decarboxylation reaction Methods 0.000 claims description 22
- BWHGMFYTDQEALD-UHFFFAOYSA-N 5-amino-2-oxopentanoic acid Chemical compound [NH3+]CCCC(=O)C([O-])=O BWHGMFYTDQEALD-UHFFFAOYSA-N 0.000 claims description 21
- 102000008214 Glutamate decarboxylase Human genes 0.000 claims description 20
- 108091022930 Glutamate decarboxylase Proteins 0.000 claims description 20
- 150000001299 aldehydes Chemical class 0.000 claims description 19
- 239000001963 growth medium Substances 0.000 claims description 19
- 101710181647 Putrescine aminotransferase Proteins 0.000 claims description 17
- 108010060511 4-Aminobutyrate Transaminase Proteins 0.000 claims description 15
- 102100035923 4-aminobutyrate aminotransferase, mitochondrial Human genes 0.000 claims description 15
- 101000950981 Bacillus subtilis (strain 168) Catabolic NAD-specific glutamate dehydrogenase RocG Proteins 0.000 claims description 14
- 102000016901 Glutamate dehydrogenase Human genes 0.000 claims description 14
- 229930195712 glutamate Natural products 0.000 claims description 14
- KDYFGRWQOYBRFD-UHFFFAOYSA-L succinate(2-) Chemical compound [O-]C(=O)CCC([O-])=O KDYFGRWQOYBRFD-UHFFFAOYSA-L 0.000 claims description 12
- AHLPHDHHMVZTML-BYPYZUCNSA-N L-Ornithine Chemical compound NCCC[C@H](N)C(O)=O AHLPHDHHMVZTML-BYPYZUCNSA-N 0.000 claims description 5
- AHLPHDHHMVZTML-UHFFFAOYSA-N Orn-delta-NH2 Natural products NCCCC(N)C(O)=O AHLPHDHHMVZTML-UHFFFAOYSA-N 0.000 claims description 5
- UTJLXEIPEHZYQJ-UHFFFAOYSA-N Ornithine Natural products OC(=O)C(C)CCCN UTJLXEIPEHZYQJ-UHFFFAOYSA-N 0.000 claims description 5
- 108090000691 Ornithine aminotransferases Proteins 0.000 claims description 5
- 102000004132 Ornithine aminotransferases Human genes 0.000 claims description 5
- 102000052812 Ornithine decarboxylases Human genes 0.000 claims description 5
- 108700005126 Ornithine decarboxylases Proteins 0.000 claims description 5
- 229960003104 ornithine Drugs 0.000 claims description 5
- 102000014898 transaminase activity proteins Human genes 0.000 claims 8
- XDAOHSVZXQBFMP-UHFFFAOYSA-N 3-hydroxy-2-oxopentanoic acid Chemical compound CCC(O)C(=O)C(O)=O XDAOHSVZXQBFMP-UHFFFAOYSA-N 0.000 claims 1
- 230000014509 gene expression Effects 0.000 abstract description 56
- SJZRECIVHVDYJC-UHFFFAOYSA-N 4-hydroxybutyric acid Chemical compound OCCCC(O)=O SJZRECIVHVDYJC-UHFFFAOYSA-N 0.000 description 273
- 229940088598 enzyme Drugs 0.000 description 213
- 108090000623 proteins and genes Proteins 0.000 description 122
- 239000000047 product Substances 0.000 description 73
- 102000004357 Transferases Human genes 0.000 description 70
- 108090000992 Transferases Proteins 0.000 description 70
- 239000000758 substrate Substances 0.000 description 62
- RGJOEKWQDUBAIZ-UHFFFAOYSA-N coenzime A Natural products OC1C(OP(O)(O)=O)C(COP(O)(=O)OP(O)(=O)OCC(C)(C)C(O)C(=O)NCCC(=O)NCCS)OC1N1C2=NC=NC(N)=C2N=C1 RGJOEKWQDUBAIZ-UHFFFAOYSA-N 0.000 description 61
- 239000005516 coenzyme A Substances 0.000 description 61
- 229940093530 coenzyme a Drugs 0.000 description 61
- KDTSHFARGAKYJN-UHFFFAOYSA-N dephosphocoenzyme A Natural products OC1C(O)C(COP(O)(=O)OP(O)(=O)OCC(C)(C)C(O)C(=O)NCCC(=O)NCCS)OC1N1C2=NC=NC(N)=C2N=C1 KDTSHFARGAKYJN-UHFFFAOYSA-N 0.000 description 61
- 230000000694 effects Effects 0.000 description 58
- 230000006696 biosynthetic metabolic pathway Effects 0.000 description 51
- 241000588724 Escherichia coli Species 0.000 description 50
- 238000006243 chemical reaction Methods 0.000 description 50
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 48
- 102000003960 Ligases Human genes 0.000 description 48
- 108090000364 Ligases Proteins 0.000 description 48
- 102000003929 Transaminases Human genes 0.000 description 48
- RGJOEKWQDUBAIZ-IBOSZNHHSA-N CoASH Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCS)O[C@H]1N1C2=NC=NC(N)=C2N=C1 RGJOEKWQDUBAIZ-IBOSZNHHSA-N 0.000 description 47
- KPGXRSRHYNQIFN-UHFFFAOYSA-N 2-oxoglutaric acid Chemical compound OC(=O)CCC(=O)C(O)=O KPGXRSRHYNQIFN-UHFFFAOYSA-N 0.000 description 41
- 102000004157 Hydrolases Human genes 0.000 description 41
- 108090000604 Hydrolases Proteins 0.000 description 41
- KDYFGRWQOYBRFD-UHFFFAOYSA-N succinic acid Chemical compound OC(=O)CCC(O)=O KDYFGRWQOYBRFD-UHFFFAOYSA-N 0.000 description 38
- YEJRWHAVMIAJKC-UHFFFAOYSA-N 4-Butyrolactone Chemical compound O=C1CCCO1 YEJRWHAVMIAJKC-UHFFFAOYSA-N 0.000 description 36
- QTBSBXVTEAMEQO-UHFFFAOYSA-N Acetic acid Chemical compound CC(O)=O QTBSBXVTEAMEQO-UHFFFAOYSA-N 0.000 description 36
- 102000005566 Succinate-Semialdehyde Dehydrogenase Human genes 0.000 description 36
- 108010084086 Succinate-Semialdehyde Dehydrogenase Proteins 0.000 description 36
- 102000004169 proteins and genes Human genes 0.000 description 36
- UKAUYVFTDYCKQA-VKHMYHEASA-N L-homoserine Chemical compound OC(=O)[C@@H](N)CCO UKAUYVFTDYCKQA-VKHMYHEASA-N 0.000 description 33
- WYURNTSHIVDZCO-UHFFFAOYSA-N Tetrahydrofuran Chemical compound C1CCOC1 WYURNTSHIVDZCO-UHFFFAOYSA-N 0.000 description 33
- 241000894007 species Species 0.000 description 33
- 235000018102 proteins Nutrition 0.000 description 32
- ZSLZBFCDCINBPY-ZSJPKINUSA-N Acetyl-CoA Natural products O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCSC(=O)C)O[C@H]1N1C2=NC=NC(N)=C2N=C1 ZSLZBFCDCINBPY-ZSJPKINUSA-N 0.000 description 31
- UKAUYVFTDYCKQA-UHFFFAOYSA-N -2-Amino-4-hydroxybutanoic acid Natural products OC(=O)C(N)CCO UKAUYVFTDYCKQA-UHFFFAOYSA-N 0.000 description 30
- 230000001419 dependent effect Effects 0.000 description 30
- 102000007698 Alcohol dehydrogenase Human genes 0.000 description 27
- 102000005369 Aldehyde Dehydrogenase Human genes 0.000 description 27
- 108020002663 Aldehyde Dehydrogenase Proteins 0.000 description 27
- 230000009615 deamination Effects 0.000 description 27
- 238000006481 deamination reaction Methods 0.000 description 27
- 230000006870 function Effects 0.000 description 27
- 239000013612 plasmid Substances 0.000 description 27
- 108010075728 Succinate-CoA Ligases Proteins 0.000 description 25
- 102000011929 Succinate-CoA Ligases Human genes 0.000 description 25
- UIUJIQZEACWQSV-UHFFFAOYSA-N succinic semialdehyde Chemical compound OC(=O)CCC=O UIUJIQZEACWQSV-UHFFFAOYSA-N 0.000 description 24
- 229940006015 4-hydroxybutyric acid Drugs 0.000 description 23
- 239000000543 intermediate Substances 0.000 description 23
- 239000002773 nucleotide Substances 0.000 description 23
- 125000003729 nucleotide group Chemical group 0.000 description 23
- 230000004048 modification Effects 0.000 description 22
- 238000012986 modification Methods 0.000 description 22
- 230000002503 metabolic effect Effects 0.000 description 21
- 108020004705 Codon Proteins 0.000 description 20
- 125000003275 alpha amino acid group Chemical group 0.000 description 20
- 239000008103 glucose Substances 0.000 description 18
- 230000026731 phosphorylation Effects 0.000 description 18
- 238000006366 phosphorylation reaction Methods 0.000 description 18
- 229940076788 pyruvate Drugs 0.000 description 18
- 239000000126 substance Substances 0.000 description 18
- BLFRQYKZFKYQLO-UHFFFAOYSA-N 4-aminobutan-1-ol Chemical compound NCCCCO BLFRQYKZFKYQLO-UHFFFAOYSA-N 0.000 description 17
- 108700024126 Butyrate kinases Proteins 0.000 description 17
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 17
- LCTONWCANYUPML-UHFFFAOYSA-M Pyruvate Chemical compound CC(=O)C([O-])=O LCTONWCANYUPML-UHFFFAOYSA-M 0.000 description 17
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 17
- WQZGKKKJIJFFOK-VFUOTHLCSA-N beta-D-glucose Chemical compound OC[C@H]1O[C@@H](O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-VFUOTHLCSA-N 0.000 description 17
- VHKNBDIQDAXGBL-UHFFFAOYSA-N 2,5-dioxopentanoic acid Chemical compound OC(=O)C(=O)CCC=O VHKNBDIQDAXGBL-UHFFFAOYSA-N 0.000 description 16
- FERIUCNNQQJTOY-UHFFFAOYSA-N Butyric acid Chemical compound CCCC(O)=O FERIUCNNQQJTOY-UHFFFAOYSA-N 0.000 description 16
- 235000014680 Saccharomyces cerevisiae Nutrition 0.000 description 16
- HWXBTNAVRSUOJR-UHFFFAOYSA-N alpha-hydroxyglutaric acid Natural products OC(=O)C(O)CCC(O)=O HWXBTNAVRSUOJR-UHFFFAOYSA-N 0.000 description 16
- 229940009533 alpha-ketoglutaric acid Drugs 0.000 description 16
- 229960003692 gamma aminobutyric acid Drugs 0.000 description 16
- 239000001384 succinic acid Substances 0.000 description 16
- 108010021809 Alcohol dehydrogenase Proteins 0.000 description 15
- 230000001851 biosynthetic effect Effects 0.000 description 15
- 239000006225 natural substrate Substances 0.000 description 15
- 241000605862 Porphyromonas gingivalis Species 0.000 description 14
- 230000001965 increasing effect Effects 0.000 description 14
- YLQBMQCUIZJEEH-UHFFFAOYSA-N tetrahydrofuran Natural products C=1C=COC=1 YLQBMQCUIZJEEH-UHFFFAOYSA-N 0.000 description 14
- HHFBTTVZSVBPFP-CITAKDKDSA-N 4-aminobutanoyl-CoA Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCSC(=O)CCCN)O[C@H]1N1C2=NC=NC(N)=C2N=C1 HHFBTTVZSVBPFP-CITAKDKDSA-N 0.000 description 13
- 102000001253 Protein Kinase Human genes 0.000 description 13
- OJFDKHTZOUZBOS-CITAKDKDSA-N acetoacetyl-CoA Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCSC(=O)CC(=O)C)O[C@H]1N1C2=NC=NC(N)=C2N=C1 OJFDKHTZOUZBOS-CITAKDKDSA-N 0.000 description 13
- 241000193401 Clostridium acetobutylicum Species 0.000 description 12
- 101710181816 Pyruvate-formate-lyase deactivase Proteins 0.000 description 12
- 239000002243 precursor Substances 0.000 description 12
- 108060006633 protein kinase Proteins 0.000 description 12
- WHUUTDBJXJRKMK-UHFFFAOYSA-N Glutamic acid Natural products OC(=O)C(N)CCC(O)=O WHUUTDBJXJRKMK-UHFFFAOYSA-N 0.000 description 11
- 238000013461 design Methods 0.000 description 11
- 238000000855 fermentation Methods 0.000 description 11
- 230000004151 fermentation Effects 0.000 description 11
- 229960002989 glutamic acid Drugs 0.000 description 11
- 235000013922 glutamic acid Nutrition 0.000 description 11
- 239000004220 glutamic acid Substances 0.000 description 11
- -1 putrescine series compounds Chemical class 0.000 description 11
- 230000001105 regulatory effect Effects 0.000 description 11
- KDVFRMMRZOCFLS-UHFFFAOYSA-N 2-oxopentanoic acid Chemical compound CCCC(=O)C(O)=O KDVFRMMRZOCFLS-UHFFFAOYSA-N 0.000 description 10
- QYHKIXYHXCJHTR-CITAKDKDSA-N 5-[2-[3-[[(2r)-4-[[[(2r,3s,4r,5r)-5-(6-aminopurin-9-yl)-4-hydroxy-3-phosphonooxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-hydroxyphosphoryl]oxy-2-hydroxy-3,3-dimethylbutanoyl]amino]propanoylamino]ethylsulfanyl]-4,5-dioxopentanoic acid Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCSC(=O)C(=O)CCC(O)=O)O[C@H]1N1C2=NC=NC(N)=C2N=C1 QYHKIXYHXCJHTR-CITAKDKDSA-N 0.000 description 10
- 241000894006 Bacteria Species 0.000 description 10
- 108091000080 Phosphotransferase Proteins 0.000 description 10
- LCTONWCANYUPML-UHFFFAOYSA-N Pyruvic acid Chemical compound CC(=O)C(O)=O LCTONWCANYUPML-UHFFFAOYSA-N 0.000 description 10
- 239000002253 acid Substances 0.000 description 10
- 239000011942 biocatalyst Substances 0.000 description 10
- 210000004027 cell Anatomy 0.000 description 10
- 230000002950 deficient Effects 0.000 description 10
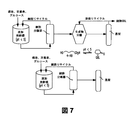
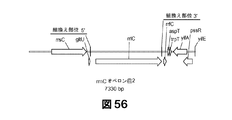
- 238000010586 diagram Methods 0.000 description 10
- 238000012239 gene modification Methods 0.000 description 10
- 230000005017 genetic modification Effects 0.000 description 10
- 235000013617 genetically modified food Nutrition 0.000 description 10
- 229940049906 glutamate Drugs 0.000 description 10
- 229930027945 nicotinamide-adenine dinucleotide Natural products 0.000 description 10
- BOPGDPNILDQYTO-NNYOXOHSSA-N nicotinamide-adenine dinucleotide Chemical compound C1=CCC(C(=O)N)=CN1[C@H]1[C@H](O)[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OC[C@@H]2[C@H]([C@@H](O)[C@@H](O2)N2C3=NC=NC(N)=C3N=C2)O)O1 BOPGDPNILDQYTO-NNYOXOHSSA-N 0.000 description 10
- 102000020233 phosphotransferase Human genes 0.000 description 10
- 238000003786 synthesis reaction Methods 0.000 description 10
- DZQLQEYLEYWJIB-UHFFFAOYSA-N 4-aminobutanal Chemical compound NCCCC=O DZQLQEYLEYWJIB-UHFFFAOYSA-N 0.000 description 9
- 101150049512 ald gene Proteins 0.000 description 9
- 230000001976 improved effect Effects 0.000 description 9
- KHPXUQMNIQBQEV-UHFFFAOYSA-N oxaloacetic acid Chemical compound OC(=O)CC(=O)C(O)=O KHPXUQMNIQBQEV-UHFFFAOYSA-N 0.000 description 9
- 229930029653 phosphoenolpyruvate Natural products 0.000 description 9
- DTBNBXWJWCWCIK-UHFFFAOYSA-K phosphonatoenolpyruvate Chemical compound [O-]C(=O)C(=C)OP([O-])([O-])=O DTBNBXWJWCWCIK-UHFFFAOYSA-K 0.000 description 9
- 229920001184 polypeptide Polymers 0.000 description 9
- 230000008569 process Effects 0.000 description 9
- 108090000765 processed proteins & peptides Proteins 0.000 description 9
- 102000004196 processed proteins & peptides Human genes 0.000 description 9
- OQEBBZSWEGYTPG-UHFFFAOYSA-N 3-aminobutanoic acid Chemical compound CC(N)CC(O)=O OQEBBZSWEGYTPG-UHFFFAOYSA-N 0.000 description 8
- 229940024606 amino acid Drugs 0.000 description 8
- QVGXLLKOCUKJST-UHFFFAOYSA-N atomic oxygen Chemical compound [O] QVGXLLKOCUKJST-UHFFFAOYSA-N 0.000 description 8
- 230000012010 growth Effects 0.000 description 8
- 230000001939 inductive effect Effects 0.000 description 8
- 230000003834 intracellular effect Effects 0.000 description 8
- 230000035772 mutation Effects 0.000 description 8
- 239000001301 oxygen Substances 0.000 description 8
- 229910052760 oxygen Inorganic materials 0.000 description 8
- 239000013598 vector Substances 0.000 description 8
- 239000002028 Biomass Substances 0.000 description 7
- CZWARROQQFCFJB-UHFFFAOYSA-N L-2-Amino-5-hydroxypentanoic acid Chemical compound OC(=O)C(N)CCCO CZWARROQQFCFJB-UHFFFAOYSA-N 0.000 description 7
- FFEARJCKVFRZRR-BYPYZUCNSA-N L-methionine Chemical compound CSCC[C@H](N)C(O)=O FFEARJCKVFRZRR-BYPYZUCNSA-N 0.000 description 7
- 235000001014 amino acid Nutrition 0.000 description 7
- 229910052799 carbon Inorganic materials 0.000 description 7
- 239000012634 fragment Substances 0.000 description 7
- 239000002609 medium Substances 0.000 description 7
- 230000037353 metabolic pathway Effects 0.000 description 7
- 238000006241 metabolic reaction Methods 0.000 description 7
- 229930182817 methionine Natural products 0.000 description 7
- 235000006109 methionine Nutrition 0.000 description 7
- 239000000376 reactant Substances 0.000 description 7
- 108010030844 2-methylcitrate synthase Proteins 0.000 description 6
- TYEYBOSBBBHJIV-UHFFFAOYSA-N 2-oxobutanoic acid Chemical compound CCC(=O)C(O)=O TYEYBOSBBBHJIV-UHFFFAOYSA-N 0.000 description 6
- RMQJECWPWQIIPW-OWOJBTEDSA-N 4-hydroxy-crotonic acid Chemical compound OC\C=C\C(O)=O RMQJECWPWQIIPW-OWOJBTEDSA-N 0.000 description 6
- ZKHQWZAMYRWXGA-KQYNXXCUSA-N Adenosine triphosphate Chemical compound C1=NC=2C(N)=NC=NC=2N1[C@@H]1O[C@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)[C@@H](O)[C@H]1O ZKHQWZAMYRWXGA-KQYNXXCUSA-N 0.000 description 6
- OKTJSMMVPCPJKN-UHFFFAOYSA-N Carbon Chemical compound [C] OKTJSMMVPCPJKN-UHFFFAOYSA-N 0.000 description 6
- 108010071536 Citrate (Si)-synthase Proteins 0.000 description 6
- 102000006732 Citrate synthase Human genes 0.000 description 6
- 108020004414 DNA Proteins 0.000 description 6
- 108020002908 Epoxide hydrolase Proteins 0.000 description 6
- OKKJLVBELUTLKV-UHFFFAOYSA-N Methanol Chemical compound OC OKKJLVBELUTLKV-UHFFFAOYSA-N 0.000 description 6
- 241000186366 Mycobacterium bovis Species 0.000 description 6
- 241000589776 Pseudomonas putida Species 0.000 description 6
- 101100208039 Rattus norvegicus Trpv5 gene Proteins 0.000 description 6
- SRBFZHDQGSBBOR-UHFFFAOYSA-N beta-D-Pyranose-Lyxose Natural products OC1COC(O)C(O)C1O SRBFZHDQGSBBOR-UHFFFAOYSA-N 0.000 description 6
- 230000004907 flux Effects 0.000 description 6
- 101150106096 gltA gene Proteins 0.000 description 6
- KWIUHFFTVRNATP-UHFFFAOYSA-N glycine betaine Chemical compound C[N+](C)(C)CC([O-])=O KWIUHFFTVRNATP-UHFFFAOYSA-N 0.000 description 6
- 101150109249 lacI gene Proteins 0.000 description 6
- 101150003321 lpdA gene Proteins 0.000 description 6
- 230000004060 metabolic process Effects 0.000 description 6
- XGAWKWCCTIEGRI-UHFFFAOYSA-N phosphono 4-hydroxybutanoate Chemical compound OCCCC(=O)OP(O)(O)=O XGAWKWCCTIEGRI-UHFFFAOYSA-N 0.000 description 6
- 230000002441 reversible effect Effects 0.000 description 6
- NQPDZGIKBAWPEJ-UHFFFAOYSA-N valeric acid Chemical compound CCCCC(O)=O NQPDZGIKBAWPEJ-UHFFFAOYSA-N 0.000 description 6
- 108030005660 3-hydroxybutyryl-CoA dehydratases Proteins 0.000 description 5
- 108010035023 4-hydroxybutyryl-CoA dehydratase Proteins 0.000 description 5
- 101150021974 Adh1 gene Proteins 0.000 description 5
- FERIUCNNQQJTOY-UHFFFAOYSA-M Butyrate Chemical compound CCCC([O-])=O FERIUCNNQQJTOY-UHFFFAOYSA-M 0.000 description 5
- 102000020018 Cystathionine gamma-Lyase Human genes 0.000 description 5
- 108010045283 Cystathionine gamma-lyase Proteins 0.000 description 5
- 102000005486 Epoxide hydrolase Human genes 0.000 description 5
- 102100034013 Gamma-glutamyl phosphate reductase Human genes 0.000 description 5
- 101710198928 Gamma-glutamyl phosphate reductase Proteins 0.000 description 5
- 102000005133 Glutamate 5-kinase Human genes 0.000 description 5
- 108010016106 Glutamate-5-semialdehyde dehydrogenase Proteins 0.000 description 5
- CKLJMWTZIZZHCS-REOHCLBHSA-N L-aspartic acid Chemical compound OC(=O)[C@@H](N)CC(O)=O CKLJMWTZIZZHCS-REOHCLBHSA-N 0.000 description 5
- KABXUUFDPUOJMW-BYPYZUCNSA-N L-glutamic 5-semialdehyde Chemical compound OC(=O)[C@@H](N)CCC=O KABXUUFDPUOJMW-BYPYZUCNSA-N 0.000 description 5
- FQFRDDVZPBFYST-CITAKDKDSA-N S-[2-[3-[[(2R)-4-[[[(2R,3S,4R,5R)-5-(6-aminopurin-9-yl)-4-hydroxy-3-phosphonooxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-hydroxyphosphoryl]oxy-2-hydroxy-3,3-dimethylbutanoyl]amino]propanoylamino]ethyl] 4-hydroxybut-2-enethioate Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCSC(=O)C=CCO)O[C@H]1N1C2=NC=NC(N)=C2N=C1 FQFRDDVZPBFYST-CITAKDKDSA-N 0.000 description 5
- 229930006000 Sucrose Natural products 0.000 description 5
- CZMRCDWAGMRECN-UGDNZRGBSA-N Sucrose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 CZMRCDWAGMRECN-UGDNZRGBSA-N 0.000 description 5
- 150000001413 amino acids Chemical class 0.000 description 5
- PYMYPHUHKUWMLA-UHFFFAOYSA-N arabinose Natural products OCC(O)C(O)C(O)C=O PYMYPHUHKUWMLA-UHFFFAOYSA-N 0.000 description 5
- CDQSJQSWAWPGKG-UHFFFAOYSA-N butane-1,1-diol Chemical compound CCCC(O)O CDQSJQSWAWPGKG-UHFFFAOYSA-N 0.000 description 5
- KDYFGRWQOYBRFD-NUQCWPJISA-N butanedioic acid Chemical compound O[14C](=O)CC[14C](O)=O KDYFGRWQOYBRFD-NUQCWPJISA-N 0.000 description 5
- 239000006227 byproduct Substances 0.000 description 5
- 238000004422 calculation algorithm Methods 0.000 description 5
- 210000000349 chromosome Anatomy 0.000 description 5
- 239000013604 expression vector Substances 0.000 description 5
- 239000000178 monomer Substances 0.000 description 5
- 230000002018 overexpression Effects 0.000 description 5
- 229920000642 polymer Polymers 0.000 description 5
- 229940107700 pyruvic acid Drugs 0.000 description 5
- 239000005720 sucrose Substances 0.000 description 5
- 230000008685 targeting Effects 0.000 description 5
- 230000004102 tricarboxylic acid cycle Effects 0.000 description 5
- AECJCRHEUUKQRT-SJOXLDEWSA-N (2S)-2-amino-4-hydroxybutanoic acid [[(2R,3S,4R,5R)-5-(6-aminopurin-9-yl)-4-hydroxy-3-phosphonooxyoxolan-2-yl]methoxy-hydroxyphosphoryl] [(3R)-3-hydroxy-2,2-dimethyl-4-oxo-4-[[3-oxo-3-(2-sulfanylethylamino)propyl]amino]butyl] hydrogen phosphate Chemical compound OC(=O)[C@@H](N)CCO.O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCS)O[C@H]1N1C2=NC=NC(N)=C2N=C1 AECJCRHEUUKQRT-SJOXLDEWSA-N 0.000 description 4
- 108010055682 3-hydroxybutyryl-CoA dehydrogenase Proteins 0.000 description 4
- 108010024655 4-hydroxybutyrate CoA-transferase Proteins 0.000 description 4
- 102000006589 Alpha-ketoglutarate dehydrogenase Human genes 0.000 description 4
- 108020004306 Alpha-ketoglutarate dehydrogenase Proteins 0.000 description 4
- 108010003415 Aspartate Aminotransferases Proteins 0.000 description 4
- 102000004625 Aspartate Aminotransferases Human genes 0.000 description 4
- 101000695175 Bacillus subtilis (strain 168) Probable phosphate butyryltransferase Proteins 0.000 description 4
- 108030002325 Carboxylate reductases Proteins 0.000 description 4
- 241000193403 Clostridium Species 0.000 description 4
- 241000186570 Clostridium kluyveri Species 0.000 description 4
- SRBFZHDQGSBBOR-IOVATXLUSA-N D-xylopyranose Chemical compound O[C@@H]1COC(O)[C@H](O)[C@H]1O SRBFZHDQGSBBOR-IOVATXLUSA-N 0.000 description 4
- 241000626621 Geobacillus Species 0.000 description 4
- 241000193385 Geobacillus stearothermophilus Species 0.000 description 4
- 241000606768 Haemophilus influenzae Species 0.000 description 4
- 102100039894 Hemoglobin subunit delta Human genes 0.000 description 4
- 241000588747 Klebsiella pneumoniae Species 0.000 description 4
- 102000003855 L-lactate dehydrogenase Human genes 0.000 description 4
- 108700023483 L-lactate dehydrogenases Proteins 0.000 description 4
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 4
- AYFVYJQAPQTCCC-GBXIJSLDSA-N L-threonine Chemical compound C[C@@H](O)[C@H](N)C(O)=O AYFVYJQAPQTCCC-GBXIJSLDSA-N 0.000 description 4
- QIVBCDIJIAJPQS-VIFPVBQESA-N L-tryptophane Chemical compound C1=CC=C2C(C[C@H](N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-VIFPVBQESA-N 0.000 description 4
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 description 4
- 108091028043 Nucleic acid sequence Proteins 0.000 description 4
- AYFVYJQAPQTCCC-UHFFFAOYSA-N Threonine Natural products CC(O)C(N)C(O)=O AYFVYJQAPQTCCC-UHFFFAOYSA-N 0.000 description 4
- 239000004473 Threonine Substances 0.000 description 4
- QIVBCDIJIAJPQS-UHFFFAOYSA-N Tryptophan Natural products C1=CC=C2C(CC(N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-UHFFFAOYSA-N 0.000 description 4
- 108010093991 Vinylacetyl-CoA Delta-isomerase Proteins 0.000 description 4
- 150000001298 alcohols Chemical class 0.000 description 4
- 229960003237 betaine Drugs 0.000 description 4
- 238000010276 construction Methods 0.000 description 4
- 230000007812 deficiency Effects 0.000 description 4
- XBDQKXXYIPTUBI-UHFFFAOYSA-N dimethylselenoniopropionate Natural products CCC(O)=O XBDQKXXYIPTUBI-UHFFFAOYSA-N 0.000 description 4
- 101150042350 gltA2 gene Proteins 0.000 description 4
- LHGVFZTZFXWLCP-UHFFFAOYSA-N guaiacol Chemical compound COC1=CC=CC=C1O LHGVFZTZFXWLCP-UHFFFAOYSA-N 0.000 description 4
- 238000000126 in silico method Methods 0.000 description 4
- KQNPFQTWMSNSAP-UHFFFAOYSA-N isobutyric acid Chemical compound CC(C)C(O)=O KQNPFQTWMSNSAP-UHFFFAOYSA-N 0.000 description 4
- JVTAAEKCZFNVCJ-UHFFFAOYSA-N lactic acid Chemical compound CC(O)C(O)=O JVTAAEKCZFNVCJ-UHFFFAOYSA-N 0.000 description 4
- 229960003136 leucine Drugs 0.000 description 4
- 235000005772 leucine Nutrition 0.000 description 4
- 230000003647 oxidation Effects 0.000 description 4
- 238000007254 oxidation reaction Methods 0.000 description 4
- ZUOUZKKEUPVFJK-UHFFFAOYSA-N phenylbenzene Natural products C1=CC=CC=C1C1=CC=CC=C1 ZUOUZKKEUPVFJK-UHFFFAOYSA-N 0.000 description 4
- UEZVMMHDMIWARA-UHFFFAOYSA-M phosphonate Chemical compound [O-]P(=O)=O UEZVMMHDMIWARA-UHFFFAOYSA-M 0.000 description 4
- 238000003752 polymerase chain reaction Methods 0.000 description 4
- 150000003839 salts Chemical group 0.000 description 4
- 238000000926 separation method Methods 0.000 description 4
- 238000002741 site-directed mutagenesis Methods 0.000 description 4
- 101150111745 sucA gene Proteins 0.000 description 4
- 101150031436 sucD gene Proteins 0.000 description 4
- 230000009466 transformation Effects 0.000 description 4
- VTEIFHQUZWABDE-UHFFFAOYSA-N 2-(2,5-dimethoxy-4-methylphenyl)-2-methoxyethanamine Chemical compound COC(CN)C1=CC(OC)=C(C)C=C1OC VTEIFHQUZWABDE-UHFFFAOYSA-N 0.000 description 3
- 108010006229 Acetyl-CoA C-acetyltransferase Proteins 0.000 description 3
- 102000005345 Acetyl-CoA C-acetyltransferase Human genes 0.000 description 3
- 244000063299 Bacillus subtilis Species 0.000 description 3
- 235000014469 Bacillus subtilis Nutrition 0.000 description 3
- UHOVQNZJYSORNB-UHFFFAOYSA-N Benzene Chemical compound C1=CC=CC=C1 UHOVQNZJYSORNB-UHFFFAOYSA-N 0.000 description 3
- 241000186312 Brevibacterium sp. Species 0.000 description 3
- WWZKQHOCKIZLMA-UHFFFAOYSA-N Caprylic acid Natural products CCCCCCCC(O)=O WWZKQHOCKIZLMA-UHFFFAOYSA-N 0.000 description 3
- 108060006006 Cytochrome-c peroxidase Proteins 0.000 description 3
- 241000196324 Embryophyta Species 0.000 description 3
- 241000282326 Felis catus Species 0.000 description 3
- WSFSSNUMVMOOMR-UHFFFAOYSA-N Formaldehyde Chemical compound O=C WSFSSNUMVMOOMR-UHFFFAOYSA-N 0.000 description 3
- 241000233866 Fungi Species 0.000 description 3
- 206010064571 Gene mutation Diseases 0.000 description 3
- QNAYBMKLOCPYGJ-REOHCLBHSA-N L-alanine Chemical compound C[C@H](N)C(O)=O QNAYBMKLOCPYGJ-REOHCLBHSA-N 0.000 description 3
- OUYCCCASQSFEME-QMMMGPOBSA-N L-tyrosine Chemical compound OC(=O)[C@@H](N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-QMMMGPOBSA-N 0.000 description 3
- 101150052992 PTBP1 gene Proteins 0.000 description 3
- 102000016387 Pancreatic elastase Human genes 0.000 description 3
- 108010067372 Pancreatic elastase Proteins 0.000 description 3
- 241000193390 Parageobacillus thermoglucosidasius Species 0.000 description 3
- 241000589614 Pseudomonas stutzeri Species 0.000 description 3
- 101100398785 Streptococcus agalactiae serotype V (strain ATCC BAA-611 / 2603 V/R) ldhD gene Proteins 0.000 description 3
- YXFVVABEGXRONW-UHFFFAOYSA-N Toluene Chemical compound CC1=CC=CC=C1 YXFVVABEGXRONW-UHFFFAOYSA-N 0.000 description 3
- 101100386830 Zymomonas mobilis subsp. mobilis (strain ATCC 31821 / ZM4 / CP4) ddh gene Proteins 0.000 description 3
- WDJHALXBUFZDSR-UHFFFAOYSA-M acetoacetate Chemical compound CC(=O)CC([O-])=O WDJHALXBUFZDSR-UHFFFAOYSA-M 0.000 description 3
- 230000002378 acidificating effect Effects 0.000 description 3
- 238000007792 addition Methods 0.000 description 3
- 229960003767 alanine Drugs 0.000 description 3
- 235000004279 alanine Nutrition 0.000 description 3
- 125000001931 aliphatic group Chemical group 0.000 description 3
- 150000004716 alpha keto acids Chemical class 0.000 description 3
- HSFWRNGVRCDJHI-UHFFFAOYSA-N alpha-acetylene Natural products C#C HSFWRNGVRCDJHI-UHFFFAOYSA-N 0.000 description 3
- 238000004458 analytical method Methods 0.000 description 3
- 101150117498 arcA gene Proteins 0.000 description 3
- 235000003704 aspartic acid Nutrition 0.000 description 3
- 230000008901 benefit Effects 0.000 description 3
- OQFSQFPPLPISGP-UHFFFAOYSA-N beta-carboxyaspartic acid Natural products OC(=O)C(N)C(C(O)=O)C(O)=O OQFSQFPPLPISGP-UHFFFAOYSA-N 0.000 description 3
- 150000001720 carbohydrates Chemical class 0.000 description 3
- 235000014633 carbohydrates Nutrition 0.000 description 3
- 125000004432 carbon atom Chemical group C* 0.000 description 3
- 230000003197 catalytic effect Effects 0.000 description 3
- 230000008859 change Effects 0.000 description 3
- 238000012217 deletion Methods 0.000 description 3
- 230000037430 deletion Effects 0.000 description 3
- 230000002255 enzymatic effect Effects 0.000 description 3
- 125000002534 ethynyl group Chemical group [H]C#C* 0.000 description 3
- 238000002474 experimental method Methods 0.000 description 3
- 108010008221 formate C-acetyltransferase Proteins 0.000 description 3
- 230000010354 integration Effects 0.000 description 3
- 101150026107 ldh1 gene Proteins 0.000 description 3
- 101150041530 ldha gene Proteins 0.000 description 3
- BJEPYKJPYRNKOW-UHFFFAOYSA-N malic acid Chemical compound OC(=O)C(O)CC(O)=O BJEPYKJPYRNKOW-UHFFFAOYSA-N 0.000 description 3
- 101150048333 ptb gene Proteins 0.000 description 3
- 238000002708 random mutagenesis Methods 0.000 description 3
- 230000002829 reductive effect Effects 0.000 description 3
- 230000000241 respiratory effect Effects 0.000 description 3
- 102200000776 rs193922753 Human genes 0.000 description 3
- 238000012216 screening Methods 0.000 description 3
- 238000002415 sodium dodecyl sulfate polyacrylamide gel electrophoresis Methods 0.000 description 3
- 239000002904 solvent Substances 0.000 description 3
- 238000006467 substitution reaction Methods 0.000 description 3
- 238000012546 transfer Methods 0.000 description 3
- OUYCCCASQSFEME-UHFFFAOYSA-N tyrosine Natural products OC(=O)C(N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-UHFFFAOYSA-N 0.000 description 3
- 235000002374 tyrosine Nutrition 0.000 description 3
- 229940005605 valeric acid Drugs 0.000 description 3
- BJEPYKJPYRNKOW-REOHCLBHSA-N (S)-malic acid Chemical compound OC(=O)[C@@H](O)CC(O)=O BJEPYKJPYRNKOW-REOHCLBHSA-N 0.000 description 2
- GEWWCWZGHNIUBW-UHFFFAOYSA-N 1-(4-nitrophenyl)propan-2-one Chemical compound CC(=O)CC1=CC=C([N+]([O-])=O)C=C1 GEWWCWZGHNIUBW-UHFFFAOYSA-N 0.000 description 2
- FGSBNBBHOZHUBO-UHFFFAOYSA-N 2-oxoadipic acid Chemical compound OC(=O)CCCC(=O)C(O)=O FGSBNBBHOZHUBO-UHFFFAOYSA-N 0.000 description 2
- XNIHZNNZJHYHLC-UHFFFAOYSA-N 2-oxohexanoic acid Chemical compound CCCCC(=O)C(O)=O XNIHZNNZJHYHLC-UHFFFAOYSA-N 0.000 description 2
- 108010046716 3-Methyl-2-Oxobutanoate Dehydrogenase (Lipoamide) Proteins 0.000 description 2
- KPULXFNPTWGJQH-UHFFFAOYSA-N 3-hydroxy-4-oxo-4-propan-2-yloxybutanoic acid Chemical compound CC(C)OC(=O)C(O)CC(O)=O KPULXFNPTWGJQH-UHFFFAOYSA-N 0.000 description 2
- QHKABHOOEWYVLI-UHFFFAOYSA-N 3-methyl-2-oxobutanoic acid Chemical compound CC(C)C(=O)C(O)=O QHKABHOOEWYVLI-UHFFFAOYSA-N 0.000 description 2
- QFVHZQCOUORWEI-UHFFFAOYSA-N 4-[(4-anilino-5-sulfonaphthalen-1-yl)diazenyl]-5-hydroxynaphthalene-2,7-disulfonic acid Chemical compound C=12C(O)=CC(S(O)(=O)=O)=CC2=CC(S(O)(=O)=O)=CC=1N=NC(C1=CC=CC(=C11)S(O)(=O)=O)=CC=C1NC1=CC=CC=C1 QFVHZQCOUORWEI-UHFFFAOYSA-N 0.000 description 2
- 101710094518 4-aminobutyrate aminotransferase Proteins 0.000 description 2
- IKHGUXGNUITLKF-UHFFFAOYSA-N Acetaldehyde Chemical compound CC=O IKHGUXGNUITLKF-UHFFFAOYSA-N 0.000 description 2
- 108030002957 Acetate CoA-transferases Proteins 0.000 description 2
- WDJHALXBUFZDSR-UHFFFAOYSA-N Acetoacetic acid Natural products CC(=O)CC(O)=O WDJHALXBUFZDSR-UHFFFAOYSA-N 0.000 description 2
- 241000193451 Acetoanaerobium sticklandii Species 0.000 description 2
- 241001165345 Acinetobacter baylyi Species 0.000 description 2
- 102000006534 Amino Acid Isomerases Human genes 0.000 description 2
- 108010008830 Amino Acid Isomerases Proteins 0.000 description 2
- 108090000673 Ammonia-Lyases Proteins 0.000 description 2
- 102000004118 Ammonia-Lyases Human genes 0.000 description 2
- 241000203069 Archaea Species 0.000 description 2
- 108700016171 Aspartate ammonia-lyases Proteins 0.000 description 2
- 241000193755 Bacillus cereus Species 0.000 description 2
- 108010071778 Benzoylformate decarboxylase Proteins 0.000 description 2
- 101100446530 Bordetella pertussis (strain Tohama I / ATCC BAA-589 / NCTC 13251) fhaE gene Proteins 0.000 description 2
- ZTQSAGDEMFDKMZ-UHFFFAOYSA-N Butyraldehyde Chemical compound CCCC=O ZTQSAGDEMFDKMZ-UHFFFAOYSA-N 0.000 description 2
- 101150116295 CAT2 gene Proteins 0.000 description 2
- 101100326920 Caenorhabditis elegans ctl-1 gene Proteins 0.000 description 2
- 241000589875 Campylobacter jejuni Species 0.000 description 2
- CURLTUGMZLYLDI-UHFFFAOYSA-N Carbon dioxide Chemical compound O=C=O CURLTUGMZLYLDI-UHFFFAOYSA-N 0.000 description 2
- 108010019670 Chimeric Antigen Receptors Proteins 0.000 description 2
- 241000193163 Clostridioides difficile Species 0.000 description 2
- 241000193454 Clostridium beijerinckii Species 0.000 description 2
- 101001060691 Clostridium sporogenes (strain ATCC 7955 / DSM 767 / NBRC 16411 / NCIMB 8053 / NCTC 8594 / PA 3679) Aromatic 2-oxoacid reductase Proteins 0.000 description 2
- 241000186520 Clostridium tetanomorphum Species 0.000 description 2
- 102000005870 Coenzyme A Ligases Human genes 0.000 description 2
- 241000186226 Corynebacterium glutamicum Species 0.000 description 2
- 241000252867 Cupriavidus metallidurans Species 0.000 description 2
- 108010048689 D-lysine 5,6-aminomutase Proteins 0.000 description 2
- WQZGKKKJIJFFOK-QTVWNMPRSA-N D-mannopyranose Chemical compound OC[C@H]1OC(O)[C@@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-QTVWNMPRSA-N 0.000 description 2
- ZAQJHHRNXZUBTE-UHFFFAOYSA-N D-threo-2-Pentulose Natural products OCC(O)C(O)C(=O)CO ZAQJHHRNXZUBTE-UHFFFAOYSA-N 0.000 description 2
- 108060002716 Exonuclease Proteins 0.000 description 2
- 229930091371 Fructose Natural products 0.000 description 2
- 239000005715 Fructose Substances 0.000 description 2
- RFSUNEUAIZKAJO-ARQDHWQXSA-N Fructose Chemical compound OC[C@H]1O[C@](O)(CO)[C@@H](O)[C@@H]1O RFSUNEUAIZKAJO-ARQDHWQXSA-N 0.000 description 2
- 241000605909 Fusobacterium Species 0.000 description 2
- 108700007698 Genetic Terminator Regions Proteins 0.000 description 2
- 241000282414 Homo sapiens Species 0.000 description 2
- 241000588749 Klebsiella oxytoca Species 0.000 description 2
- 201000008225 Klebsiella pneumonia Diseases 0.000 description 2
- 241001138401 Kluyveromyces lactis Species 0.000 description 2
- KDXKERNSBIXSRK-YFKPBYRVSA-N L-lysine Chemical compound NCCCC[C@H](N)C(O)=O KDXKERNSBIXSRK-YFKPBYRVSA-N 0.000 description 2
- COLNVLDHVKWLRT-QMMMGPOBSA-N L-phenylalanine Chemical compound OC(=O)[C@@H](N)CC1=CC=CC=C1 COLNVLDHVKWLRT-QMMMGPOBSA-N 0.000 description 2
- KZSNJWFQEVHDMF-BYPYZUCNSA-N L-valine Chemical compound CC(C)[C@H](N)C(O)=O KZSNJWFQEVHDMF-BYPYZUCNSA-N 0.000 description 2
- 241000235087 Lachancea kluyveri Species 0.000 description 2
- 108090001060 Lipase Proteins 0.000 description 2
- 102000004882 Lipase Human genes 0.000 description 2
- 239000004367 Lipase Substances 0.000 description 2
- 108010011449 Long-chain-fatty-acid-CoA ligase Proteins 0.000 description 2
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 description 2
- 239000004472 Lysine Substances 0.000 description 2
- 101150058595 MDH gene Proteins 0.000 description 2
- 108010026217 Malate Dehydrogenase Proteins 0.000 description 2
- 102000013460 Malate Dehydrogenase Human genes 0.000 description 2
- 102000006833 Multifunctional Enzymes Human genes 0.000 description 2
- 108010047290 Multifunctional Enzymes Proteins 0.000 description 2
- 241000186367 Mycobacterium avium Species 0.000 description 2
- 241001607431 Mycobacterium marinum M Species 0.000 description 2
- 241000187479 Mycobacterium tuberculosis Species 0.000 description 2
- 101100126846 Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) katG gene Proteins 0.000 description 2
- 241001197104 Nocardia iowensis Species 0.000 description 2
- 229910019142 PO4 Inorganic materials 0.000 description 2
- 208000026681 Paratuberculosis Diseases 0.000 description 2
- 206010035717 Pneumonia klebsiella Diseases 0.000 description 2
- 241000986839 Porphyromonas gingivalis W83 Species 0.000 description 2
- 241000589516 Pseudomonas Species 0.000 description 2
- 241000589540 Pseudomonas fluorescens Species 0.000 description 2
- 108010011939 Pyruvate Decarboxylase Proteins 0.000 description 2
- 241000235347 Schizosaccharomyces pombe Species 0.000 description 2
- 102000012479 Serine Proteases Human genes 0.000 description 2
- 108010022999 Serine Proteases Proteins 0.000 description 2
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 2
- 229920002472 Starch Polymers 0.000 description 2
- 244000057717 Streptococcus lactis Species 0.000 description 2
- 235000014897 Streptococcus lactis Nutrition 0.000 description 2
- QAOWNCQODCNURD-UHFFFAOYSA-N Sulfuric acid Chemical compound OS(O)(=O)=O QAOWNCQODCNURD-UHFFFAOYSA-N 0.000 description 2
- 101000716799 Sus scrofa Succinyl-CoA:3-ketoacid coenzyme A transferase 1, mitochondrial Proteins 0.000 description 2
- 241000589499 Thermus thermophilus Species 0.000 description 2
- 102100033451 Thyroid hormone receptor beta Human genes 0.000 description 2
- 241000223105 Trypanosoma brucei Species 0.000 description 2
- KZSNJWFQEVHDMF-UHFFFAOYSA-N Valine Natural products CC(C)C(N)C(O)=O KZSNJWFQEVHDMF-UHFFFAOYSA-N 0.000 description 2
- 241000588902 Zymomonas mobilis Species 0.000 description 2
- 238000009825 accumulation Methods 0.000 description 2
- 101150015189 aceE gene Proteins 0.000 description 2
- 125000000218 acetic acid group Chemical group C(C)(=O)* 0.000 description 2
- 125000002252 acyl group Chemical group 0.000 description 2
- 230000003044 adaptive effect Effects 0.000 description 2
- PYMYPHUHKUWMLA-LMVFSUKVSA-N aldehydo-D-ribose Chemical compound OC[C@@H](O)[C@@H](O)[C@@H](O)C=O PYMYPHUHKUWMLA-LMVFSUKVSA-N 0.000 description 2
- WQZGKKKJIJFFOK-PHYPRBDBSA-N alpha-D-galactose Chemical compound OC[C@H]1O[C@H](O)[C@H](O)[C@@H](O)[C@H]1O WQZGKKKJIJFFOK-PHYPRBDBSA-N 0.000 description 2
- 125000000539 amino acid group Chemical group 0.000 description 2
- QGZKDVFQNNGYKY-UHFFFAOYSA-O ammonium group Chemical group [NH4+] QGZKDVFQNNGYKY-UHFFFAOYSA-O 0.000 description 2
- 230000003321 amplification Effects 0.000 description 2
- 238000013459 approach Methods 0.000 description 2
- PYMYPHUHKUWMLA-WDCZJNDASA-N arabinose Chemical compound OC[C@@H](O)[C@@H](O)[C@H](O)C=O PYMYPHUHKUWMLA-WDCZJNDASA-N 0.000 description 2
- 229940009098 aspartate Drugs 0.000 description 2
- 239000012298 atmosphere Substances 0.000 description 2
- 230000001580 bacterial effect Effects 0.000 description 2
- 230000008238 biochemical pathway Effects 0.000 description 2
- 238000005842 biochemical reaction Methods 0.000 description 2
- 239000004305 biphenyl Substances 0.000 description 2
- 235000010290 biphenyl Nutrition 0.000 description 2
- 150000005693 branched-chain amino acids Chemical class 0.000 description 2
- 101150031417 buk1 gene Proteins 0.000 description 2
- 229930188620 butyrolactone Natural products 0.000 description 2
- 125000003178 carboxy group Chemical group [H]OC(*)=O 0.000 description 2
- 230000015556 catabolic process Effects 0.000 description 2
- 238000009903 catalytic hydrogenation reaction Methods 0.000 description 2
- 239000003795 chemical substances by application Substances 0.000 description 2
- 230000002759 chromosomal effect Effects 0.000 description 2
- 230000000295 complement effect Effects 0.000 description 2
- 239000013078 crystal Substances 0.000 description 2
- 230000007423 decrease Effects 0.000 description 2
- 230000003247 decreasing effect Effects 0.000 description 2
- 238000006731 degradation reaction Methods 0.000 description 2
- WOWBFOBYOAGEEA-UHFFFAOYSA-N diafenthiuron Chemical compound CC(C)C1=C(NC(=S)NC(C)(C)C)C(C(C)C)=CC(OC=2C=CC=CC=2)=C1 WOWBFOBYOAGEEA-UHFFFAOYSA-N 0.000 description 2
- 239000010432 diamond Substances 0.000 description 2
- 230000009088 enzymatic function Effects 0.000 description 2
- 210000003527 eukaryotic cell Anatomy 0.000 description 2
- 102000013165 exonuclease Human genes 0.000 description 2
- 101150093898 fimD gene Proteins 0.000 description 2
- 229930182830 galactose Natural products 0.000 description 2
- 230000007614 genetic variation Effects 0.000 description 2
- 229960001867 guaiacol Drugs 0.000 description 2
- 229940047650 haemophilus influenzae Drugs 0.000 description 2
- MNWFXJYAOYHMED-UHFFFAOYSA-N heptanoic acid Chemical compound CCCCCCC(O)=O MNWFXJYAOYHMED-UHFFFAOYSA-N 0.000 description 2
- 238000004128 high performance liquid chromatography Methods 0.000 description 2
- 108010071598 homoserine kinase Proteins 0.000 description 2
- 150000001261 hydroxy acids Chemical class 0.000 description 2
- 230000006872 improvement Effects 0.000 description 2
- 238000000338 in vitro Methods 0.000 description 2
- 238000003780 insertion Methods 0.000 description 2
- 230000037431 insertion Effects 0.000 description 2
- FGKJLKRYENPLQH-UHFFFAOYSA-N isocaproic acid Chemical compound CC(C)CCC(O)=O FGKJLKRYENPLQH-UHFFFAOYSA-N 0.000 description 2
- 238000006317 isomerization reaction Methods 0.000 description 2
- 150000004715 keto acids Chemical class 0.000 description 2
- BTNMPGBKDVTSJY-UHFFFAOYSA-N keto-phenylpyruvic acid Chemical compound OC(=O)C(=O)CC1=CC=CC=C1 BTNMPGBKDVTSJY-UHFFFAOYSA-N 0.000 description 2
- 239000004310 lactic acid Substances 0.000 description 2
- 235000014655 lactic acid Nutrition 0.000 description 2
- 235000019421 lipase Nutrition 0.000 description 2
- 239000007788 liquid Substances 0.000 description 2
- 235000018977 lysine Nutrition 0.000 description 2
- 238000012423 maintenance Methods 0.000 description 2
- FPYJFEHAWHCUMM-UHFFFAOYSA-N maleic anhydride Chemical compound O=C1OC(=O)C=C1 FPYJFEHAWHCUMM-UHFFFAOYSA-N 0.000 description 2
- 239000001630 malic acid Substances 0.000 description 2
- 235000011090 malic acid Nutrition 0.000 description 2
- 239000003550 marker Substances 0.000 description 2
- 239000002207 metabolite Substances 0.000 description 2
- 210000003470 mitochondria Anatomy 0.000 description 2
- 230000002438 mitochondrial effect Effects 0.000 description 2
- 231100000350 mutagenesis Toxicity 0.000 description 2
- 238000002703 mutagenesis Methods 0.000 description 2
- 238000003199 nucleic acid amplification method Methods 0.000 description 2
- 235000015097 nutrients Nutrition 0.000 description 2
- 230000003204 osmotic effect Effects 0.000 description 2
- 230000008723 osmotic stress Effects 0.000 description 2
- 125000005522 oxopentanoic acid group Chemical group 0.000 description 2
- 101150111581 pflB gene Proteins 0.000 description 2
- 239000012071 phase Substances 0.000 description 2
- LCPDWSOZIOUXRV-UHFFFAOYSA-N phenoxyacetic acid Chemical compound OC(=O)COC1=CC=CC=C1 LCPDWSOZIOUXRV-UHFFFAOYSA-N 0.000 description 2
- WLJVXDMOQOGPHL-UHFFFAOYSA-N phenylacetic acid Chemical compound OC(=O)CC1=CC=CC=C1 WLJVXDMOQOGPHL-UHFFFAOYSA-N 0.000 description 2
- COLNVLDHVKWLRT-UHFFFAOYSA-N phenylalanine Natural products OC(=O)C(N)CC1=CC=CC=C1 COLNVLDHVKWLRT-UHFFFAOYSA-N 0.000 description 2
- 235000008729 phenylalanine Nutrition 0.000 description 2
- 239000010452 phosphate Substances 0.000 description 2
- 210000001236 prokaryotic cell Anatomy 0.000 description 2
- 235000019260 propionic acid Nutrition 0.000 description 2
- 230000004952 protein activity Effects 0.000 description 2
- 230000017854 proteolysis Effects 0.000 description 2
- IUVKMZGDUIUOCP-BTNSXGMBSA-N quinbolone Chemical compound O([C@H]1CC[C@H]2[C@H]3[C@@H]([C@]4(C=CC(=O)C=C4CC3)C)CC[C@@]21C)C1=CCCC1 IUVKMZGDUIUOCP-BTNSXGMBSA-N 0.000 description 2
- 230000006798 recombination Effects 0.000 description 2
- 230000009467 reduction Effects 0.000 description 2
- 238000006722 reduction reaction Methods 0.000 description 2
- 230000028327 secretion Effects 0.000 description 2
- 238000002864 sequence alignment Methods 0.000 description 2
- 239000007787 solid Substances 0.000 description 2
- 230000002269 spontaneous effect Effects 0.000 description 2
- 235000019698 starch Nutrition 0.000 description 2
- 239000008107 starch Substances 0.000 description 2
- 235000000346 sugar Nutrition 0.000 description 2
- 238000012360 testing method Methods 0.000 description 2
- 238000013518 transcription Methods 0.000 description 2
- 230000035897 transcription Effects 0.000 description 2
- 238000011144 upstream manufacturing Methods 0.000 description 2
- 239000004474 valine Substances 0.000 description 2
- 229960004295 valine Drugs 0.000 description 2
- HDTRYLNUVZCQOY-UHFFFAOYSA-N α-D-glucopyranosyl-α-D-glucopyranoside Natural products OC1C(O)C(O)C(CO)OC1OC1C(O)C(O)C(O)C(CO)O1 HDTRYLNUVZCQOY-UHFFFAOYSA-N 0.000 description 1
- PADQINQHPQKXNL-LSDHHAIUSA-N (+)-dihydrokaempferol Chemical compound C1([C@@H]2[C@H](C(C3=C(O)C=C(O)C=C3O2)=O)O)=CC=C(O)C=C1 PADQINQHPQKXNL-LSDHHAIUSA-N 0.000 description 1
- MTCFGRXMJLQNBG-REOHCLBHSA-N (2S)-2-Amino-3-hydroxypropansäure Chemical compound OC[C@H](N)C(O)=O MTCFGRXMJLQNBG-REOHCLBHSA-N 0.000 description 1
- TVYAJJQTTAXNFA-UHFFFAOYSA-N (4-nitrophenyl) 2-methyldecanoate Chemical compound CCCCCCCCC(C)C(=O)OC1=CC=C([N+]([O-])=O)C=C1 TVYAJJQTTAXNFA-UHFFFAOYSA-N 0.000 description 1
- FQVLRGLGWNWPSS-BXBUPLCLSA-N (4r,7s,10s,13s,16r)-16-acetamido-13-(1h-imidazol-5-ylmethyl)-10-methyl-6,9,12,15-tetraoxo-7-propan-2-yl-1,2-dithia-5,8,11,14-tetrazacycloheptadecane-4-carboxamide Chemical compound N1C(=O)[C@@H](NC(C)=O)CSSC[C@@H](C(N)=O)NC(=O)[C@H](C(C)C)NC(=O)[C@H](C)NC(=O)[C@@H]1CC1=CN=CN1 FQVLRGLGWNWPSS-BXBUPLCLSA-N 0.000 description 1
- PHIQHXFUZVPYII-ZCFIWIBFSA-N (R)-carnitine Chemical compound C[N+](C)(C)C[C@H](O)CC([O-])=O PHIQHXFUZVPYII-ZCFIWIBFSA-N 0.000 description 1
- PSBDWGZCVUAZQS-UHFFFAOYSA-N (dimethylsulfonio)acetate Chemical compound C[S+](C)CC([O-])=O PSBDWGZCVUAZQS-UHFFFAOYSA-N 0.000 description 1
- OZHIYEINSCNALY-UHFFFAOYSA-N 1-aminobutan-1-ol Chemical compound CCCC(N)O OZHIYEINSCNALY-UHFFFAOYSA-N 0.000 description 1
- YQMXOIAIYXXXEE-UHFFFAOYSA-N 1-benzylpyrrolidin-3-ol Chemical compound C1C(O)CCN1CC1=CC=CC=C1 YQMXOIAIYXXXEE-UHFFFAOYSA-N 0.000 description 1
- YNXICDMQCQPQEW-UHFFFAOYSA-N 1-naphthyl dihydrogen phosphate Chemical compound C1=CC=C2C(OP(O)(=O)O)=CC=CC2=C1 YNXICDMQCQPQEW-UHFFFAOYSA-N 0.000 description 1
- PKAUICCNAWQPAU-UHFFFAOYSA-N 2-(4-chloro-2-methylphenoxy)acetic acid;n-methylmethanamine Chemical compound CNC.CC1=CC(Cl)=CC=C1OCC(O)=O PKAUICCNAWQPAU-UHFFFAOYSA-N 0.000 description 1
- 108090000168 2-Oxoisovalerate Dehydrogenase (Acylating) Proteins 0.000 description 1
- HAMZPFLPCUODHG-UHFFFAOYSA-N 2-cyclopropyl-1,3-dioxane-4,6-dione Chemical compound C1(CC(=O)OC(C2CC2)O1)=O HAMZPFLPCUODHG-UHFFFAOYSA-N 0.000 description 1
- UIKQNMXWCYQNCS-UHFFFAOYSA-N 2-hydroxybutanal Chemical compound CCC(O)C=O UIKQNMXWCYQNCS-UHFFFAOYSA-N 0.000 description 1
- 239000001903 2-oxo-3-phenylpropanoic acid Substances 0.000 description 1
- WWYDYZMNFQIYPT-UHFFFAOYSA-L 2-phenylpropanedioate Chemical compound [O-]C(=O)C(C([O-])=O)C1=CC=CC=C1 WWYDYZMNFQIYPT-UHFFFAOYSA-L 0.000 description 1
- BOOCRBBDEWUMEY-UHFFFAOYSA-N 3-cyclobutyloxy-3-oxopropanoic acid Chemical compound C1CC(C1)OC(=O)CC(=O)O BOOCRBBDEWUMEY-UHFFFAOYSA-N 0.000 description 1
- WGKORGIPQUAKOI-UHFFFAOYSA-N 3-cyclopropyloxy-3-oxopropanoic acid Chemical compound OC(=O)CC(=O)OC1CC1 WGKORGIPQUAKOI-UHFFFAOYSA-N 0.000 description 1
- 101710186512 3-ketoacyl-CoA thiolase Proteins 0.000 description 1
- 239000001388 3-methyl-2-oxobutanoic acid Substances 0.000 description 1
- JJKMIZGENPMJRC-UHFFFAOYSA-N 3-oxo-3-propan-2-yloxypropanoic acid Chemical compound CC(C)OC(=O)CC(O)=O JJKMIZGENPMJRC-UHFFFAOYSA-N 0.000 description 1
- HDXXKLJVUKAUHH-UHFFFAOYSA-N 3-oxo-3-propoxypropanoic acid Chemical compound CCCOC(=O)CC(O)=O HDXXKLJVUKAUHH-UHFFFAOYSA-N 0.000 description 1
- XZKIHKMTEMTJQX-UHFFFAOYSA-N 4-Nitrophenyl Phosphate Chemical compound OP(O)(=O)OC1=CC=C([N+]([O-])=O)C=C1 XZKIHKMTEMTJQX-UHFFFAOYSA-N 0.000 description 1
- CNXHSMPAOQXDRE-BLPRJPCASA-N 4-aminobutanoic acid;[[(2r,3s,4r,5r)-5-(6-aminopurin-9-yl)-4-hydroxy-3-phosphonooxyoxolan-2-yl]methoxy-hydroxyphosphoryl] [(3r)-3-hydroxy-2,2-dimethyl-4-oxo-4-[[3-oxo-3-(2-sulfanylethylamino)propyl]amino]butyl] hydrogen phosphate Chemical group NCCCC(O)=O.O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCS)O[C@H]1N1C2=NC=NC(N)=C2N=C1 CNXHSMPAOQXDRE-BLPRJPCASA-N 0.000 description 1
- HLOFWGGVFLUZMZ-UHFFFAOYSA-N 4-hydroxy-4-(6-methoxynaphthalen-2-yl)butan-2-one Chemical compound C1=C(C(O)CC(C)=O)C=CC2=CC(OC)=CC=C21 HLOFWGGVFLUZMZ-UHFFFAOYSA-N 0.000 description 1
- SXIFAEWFOJETOA-UHFFFAOYSA-N 4-hydroxy-butyl Chemical group [CH2]CCCO SXIFAEWFOJETOA-UHFFFAOYSA-N 0.000 description 1
- KTDASVGSEYLAIG-UHFFFAOYSA-N 4-hydroxybutanal Chemical compound OCCCC=O.OCCCC=O KTDASVGSEYLAIG-UHFFFAOYSA-N 0.000 description 1
- VTHKLCWOLNQWHB-UHFFFAOYSA-N 5-aminopentan-2-one Chemical compound CC(=O)CCCN VTHKLCWOLNQWHB-UHFFFAOYSA-N 0.000 description 1
- JSHPTIGHEWEXRW-UHFFFAOYSA-N 5-hydroxypentan-2-one Chemical compound CC(=O)CCCO JSHPTIGHEWEXRW-UHFFFAOYSA-N 0.000 description 1
- QUKRTJQSGPLQKQ-UHFFFAOYSA-N 5-methylsulfonyl-3h-1,3-benzoxazol-2-one Chemical compound CS(=O)(=O)C1=CC=C2OC(=O)NC2=C1 QUKRTJQSGPLQKQ-UHFFFAOYSA-N 0.000 description 1
- QTBSBXVTEAMEQO-UHFFFAOYSA-M Acetate Chemical compound CC([O-])=O QTBSBXVTEAMEQO-UHFFFAOYSA-M 0.000 description 1
- 241000589220 Acetobacter Species 0.000 description 1
- 101100001031 Acetobacter aceti adhA gene Proteins 0.000 description 1
- 241001453369 Achromobacter denitrificans Species 0.000 description 1
- 241000604451 Acidaminococcus Species 0.000 description 1
- 241000588626 Acinetobacter baumannii Species 0.000 description 1
- 241000948980 Actinobacillus succinogenes Species 0.000 description 1
- 241000567139 Aeropyrum pernix Species 0.000 description 1
- 229920001817 Agar Polymers 0.000 description 1
- 241000589155 Agrobacterium tumefaciens Species 0.000 description 1
- 241000722954 Anaerobiospirillum succiniciproducens Species 0.000 description 1
- 241001136167 Anaerotignum propionicum Species 0.000 description 1
- 241000428313 Anaerotruncus colihominis Species 0.000 description 1
- 108010006591 Apoenzymes Proteins 0.000 description 1
- 241000219195 Arabidopsis thaliana Species 0.000 description 1
- 241000180579 Arca Species 0.000 description 1
- 241000205046 Archaeoglobus Species 0.000 description 1
- 239000004475 Arginine Substances 0.000 description 1
- 102100032948 Aspartoacylase Human genes 0.000 description 1
- 241000228245 Aspergillus niger Species 0.000 description 1
- 241001465318 Aspergillus terreus Species 0.000 description 1
- 241000194103 Bacillus pumilus Species 0.000 description 1
- 108010029692 Bisphosphoglycerate mutase Proteins 0.000 description 1
- 241000283690 Bos taurus Species 0.000 description 1
- 241000589174 Bradyrhizobium japonicum Species 0.000 description 1
- 241000193764 Brevibacillus brevis Species 0.000 description 1
- 241000589513 Burkholderia cepacia Species 0.000 description 1
- IERHLVCPSMICTF-XVFCMESISA-N CMP group Chemical group P(=O)(O)(O)OC[C@@H]1[C@H]([C@H]([C@@H](O1)N1C(=O)N=C(N)C=C1)O)O IERHLVCPSMICTF-XVFCMESISA-N 0.000 description 1
- 241000244203 Caenorhabditis elegans Species 0.000 description 1
- 241000222120 Candida <Saccharomycetales> Species 0.000 description 1
- UGFAIRIUMAVXCW-UHFFFAOYSA-N Carbon monoxide Chemical compound [O+]#[C-] UGFAIRIUMAVXCW-UHFFFAOYSA-N 0.000 description 1
- 241000192731 Chloroflexus aurantiacus Species 0.000 description 1
- 241001494522 Citrobacter amalonaticus Species 0.000 description 1
- 241001112695 Clostridiales Species 0.000 description 1
- 241001468165 Clostridium aminobutyricum Species 0.000 description 1
- 241000193468 Clostridium perfringens Species 0.000 description 1
- 241001508458 Clostridium saccharoperbutylacetonicum Species 0.000 description 1
- 241000186524 Clostridium subterminale Species 0.000 description 1
- 241000193452 Clostridium tyrobutyricum Species 0.000 description 1
- 108091026890 Coding region Proteins 0.000 description 1
- 108020004635 Complementary DNA Proteins 0.000 description 1
- 241000414116 Cyanobium Species 0.000 description 1
- 102100030497 Cytochrome c Human genes 0.000 description 1
- 108010075031 Cytochromes c Proteins 0.000 description 1
- GSXOAOHZAIYLCY-UHFFFAOYSA-N D-F6P Natural products OCC(=O)C(O)C(O)C(O)COP(O)(O)=O GSXOAOHZAIYLCY-UHFFFAOYSA-N 0.000 description 1
- 229930182843 D-Lactic acid Natural products 0.000 description 1
- WQZGKKKJIJFFOK-IVMDWMLBSA-N D-allopyranose Chemical compound OC[C@H]1OC(O)[C@H](O)[C@H](O)[C@@H]1O WQZGKKKJIJFFOK-IVMDWMLBSA-N 0.000 description 1
- JVTAAEKCZFNVCJ-UWTATZPHSA-N D-lactic acid Chemical compound C[C@@H](O)C(O)=O JVTAAEKCZFNVCJ-UWTATZPHSA-N 0.000 description 1
- SHZGCJCMOBCMKK-UHFFFAOYSA-N D-mannomethylose Natural products CC1OC(O)C(O)C(O)C1O SHZGCJCMOBCMKK-UHFFFAOYSA-N 0.000 description 1
- BJHIKXHVCXFQLS-PUFIMZNGSA-N D-psicose Chemical compound OC[C@@H](O)[C@@H](O)[C@@H](O)C(=O)CO BJHIKXHVCXFQLS-PUFIMZNGSA-N 0.000 description 1
- ZAQJHHRNXZUBTE-NQXXGFSBSA-N D-ribulose Chemical compound OC[C@@H](O)[C@@H](O)C(=O)CO ZAQJHHRNXZUBTE-NQXXGFSBSA-N 0.000 description 1
- 102000007528 DNA Polymerase III Human genes 0.000 description 1
- 108010071146 DNA Polymerase III Proteins 0.000 description 1
- 108010014303 DNA-directed DNA polymerase Proteins 0.000 description 1
- 102000016928 DNA-directed DNA polymerase Human genes 0.000 description 1
- 241001053771 Dictyostelium discoideum AX4 Species 0.000 description 1
- 108010044229 Dihydroflavanol 4-reductase Proteins 0.000 description 1
- 102000016680 Dioxygenases Human genes 0.000 description 1
- 108010028143 Dioxygenases Proteins 0.000 description 1
- 241000255581 Drosophila <fruit fly, genus> Species 0.000 description 1
- WQXNXVUDBPYKBA-UHFFFAOYSA-N Ectoine Natural products CC1=NCCC(C(O)=O)N1 WQXNXVUDBPYKBA-UHFFFAOYSA-N 0.000 description 1
- 241000305071 Enterobacterales Species 0.000 description 1
- 241000190844 Erythrobacter Species 0.000 description 1
- 108700039887 Essential Genes Proteins 0.000 description 1
- 241000193456 Eubacterium barkeri Species 0.000 description 1
- 108010046335 Ferredoxin-NADP Reductase Proteins 0.000 description 1
- 102000002464 Galactosidases Human genes 0.000 description 1
- 108010093031 Galactosidases Proteins 0.000 description 1
- 241000589232 Gluconobacter oxydans Species 0.000 description 1
- 101710150242 Glutamate decarboxylase A Proteins 0.000 description 1
- 241000204946 Halobacterium salinarum Species 0.000 description 1
- 241000204988 Haloferax mediterranei Species 0.000 description 1
- 241000589989 Helicobacter Species 0.000 description 1
- 241000238631 Hexapoda Species 0.000 description 1
- 241000282412 Homo Species 0.000 description 1
- 101000797251 Homo sapiens Aspartoacylase Proteins 0.000 description 1
- 108010001336 Horseradish Peroxidase Proteins 0.000 description 1
- DGAQECJNVWCQMB-PUAWFVPOSA-M Ilexoside XXIX Chemical compound C[C@@H]1CC[C@@]2(CC[C@@]3(C(=CC[C@H]4[C@]3(CC[C@@H]5[C@@]4(CC[C@@H](C5(C)C)OS(=O)(=O)[O-])C)C)[C@@H]2[C@]1(C)O)C)C(=O)O[C@H]6[C@@H]([C@H]([C@@H]([C@H](O6)CO)O)O)O.[Na+] DGAQECJNVWCQMB-PUAWFVPOSA-M 0.000 description 1
- 108010075869 Isocitrate Dehydrogenase Proteins 0.000 description 1
- 102000012011 Isocitrate Dehydrogenase Human genes 0.000 description 1
- 102000004195 Isomerases Human genes 0.000 description 1
- 108090000769 Isomerases Proteins 0.000 description 1
- 208000007976 Ketosis Diseases 0.000 description 1
- 241000588915 Klebsiella aerogenes Species 0.000 description 1
- 235000014663 Kluyveromyces fragilis Nutrition 0.000 description 1
- 241000235058 Komagataella pastoris Species 0.000 description 1
- XUJNEKJLAYXESH-REOHCLBHSA-N L-Cysteine Chemical compound SC[C@H](N)C(O)=O XUJNEKJLAYXESH-REOHCLBHSA-N 0.000 description 1
- ODKSFYDXXFIFQN-BYPYZUCNSA-P L-argininium(2+) Chemical compound NC(=[NH2+])NCCC[C@H]([NH3+])C(O)=O ODKSFYDXXFIFQN-BYPYZUCNSA-P 0.000 description 1
- LKDRXBCSQODPBY-NSHGFSBMSA-N L-fructose Chemical compound OCC1(O)OC[C@H](O)[C@H](O)[C@H]1O LKDRXBCSQODPBY-NSHGFSBMSA-N 0.000 description 1
- HNDVDQJCIGZPNO-YFKPBYRVSA-N L-histidine Chemical compound OC(=O)[C@@H](N)CC1=CN=CN1 HNDVDQJCIGZPNO-YFKPBYRVSA-N 0.000 description 1
- AGPKZVBTJJNPAG-WHFBIAKZSA-N L-isoleucine Chemical compound CC[C@H](C)[C@H](N)C(O)=O AGPKZVBTJJNPAG-WHFBIAKZSA-N 0.000 description 1
- WQZGKKKJIJFFOK-JFNONXLTSA-N L-mannopyranose Chemical compound OC[C@@H]1OC(O)[C@H](O)[C@H](O)[C@H]1O WQZGKKKJIJFFOK-JFNONXLTSA-N 0.000 description 1
- HXEACLLIILLPRG-YFKPBYRVSA-N L-pipecolic acid Chemical compound [O-]C(=O)[C@@H]1CCCC[NH2+]1 HXEACLLIILLPRG-YFKPBYRVSA-N 0.000 description 1
- PNNNRSAQSRJVSB-UHFFFAOYSA-N L-rhamnose Natural products CC(O)C(O)C(O)C(O)C=O PNNNRSAQSRJVSB-UHFFFAOYSA-N 0.000 description 1
- 108010001469 L-rhamnose isomerase Proteins 0.000 description 1
- QZNPNKJXABGCRC-FUTKDDECSA-N L-rhamnulose Chemical compound C[C@H](O)[C@H](O)[C@@H](O)C(=O)CO QZNPNKJXABGCRC-FUTKDDECSA-N 0.000 description 1
- ZAQJHHRNXZUBTE-WVZVXSGGSA-N L-xylulose Chemical compound OC[C@H](O)[C@@H](O)C(=O)CO ZAQJHHRNXZUBTE-WVZVXSGGSA-N 0.000 description 1
- 240000006024 Lactobacillus plantarum Species 0.000 description 1
- 235000013965 Lactobacillus plantarum Nutrition 0.000 description 1
- 241000194036 Lactococcus Species 0.000 description 1
- 241000222734 Leishmania mexicana Species 0.000 description 1
- 241000192130 Leuconostoc mesenteroides Species 0.000 description 1
- 102100025357 Lipid-phosphate phosphatase Human genes 0.000 description 1
- 108060001084 Luciferase Proteins 0.000 description 1
- 241001539803 Magnusiomyces capitatus Species 0.000 description 1
- 102100026665 Malonate-CoA ligase ACSF3, mitochondrial Human genes 0.000 description 1
- 241000124008 Mammalia Species 0.000 description 1
- 241001465754 Metazoa Species 0.000 description 1
- 102000008109 Mixed Function Oxygenases Human genes 0.000 description 1
- 108010074633 Mixed Function Oxygenases Proteins 0.000 description 1
- 241000193459 Moorella thermoacetica Species 0.000 description 1
- 241000699666 Mus <mouse, genus> Species 0.000 description 1
- 241000699660 Mus musculus Species 0.000 description 1
- 101100463018 Mus musculus Pck1 gene Proteins 0.000 description 1
- 241000187480 Mycobacterium smegmatis Species 0.000 description 1
- 241001025881 Mycobacterium smegmatis str. MC2 155 Species 0.000 description 1
- 241000204031 Mycoplasma Species 0.000 description 1
- 241000863422 Myxococcus xanthus Species 0.000 description 1
- LRHPLDYGYMQRHN-UHFFFAOYSA-N N-Butanol Chemical compound CCCCO LRHPLDYGYMQRHN-UHFFFAOYSA-N 0.000 description 1
- SECXISVLQFMRJM-UHFFFAOYSA-N N-Methylpyrrolidone Chemical compound CN1CCCC1=O SECXISVLQFMRJM-UHFFFAOYSA-N 0.000 description 1
- 241000167284 Natranaerobius Species 0.000 description 1
- 241001495644 Nicotiana glutinosa Species 0.000 description 1
- 241001503673 Nocardia farcinica Species 0.000 description 1
- 238000000636 Northern blotting Methods 0.000 description 1
- FXDNYOANAXWZHG-VKHMYHEASA-N O-phospho-L-homoserine Chemical compound OC(=O)[C@@H](N)CCOP(O)(O)=O FXDNYOANAXWZHG-VKHMYHEASA-N 0.000 description 1
- TYBWABJIIOVYOR-UHFFFAOYSA-N OCC(C(O)=O)OP(=O)=O Chemical class OCC(C(O)=O)OP(=O)=O TYBWABJIIOVYOR-UHFFFAOYSA-N 0.000 description 1
- 101710164401 Omega-aminotransferase Proteins 0.000 description 1
- 241000283973 Oryctolagus cuniculus Species 0.000 description 1
- 240000007594 Oryza sativa Species 0.000 description 1
- 235000007164 Oryza sativa Nutrition 0.000 description 1
- 108090000417 Oxygenases Proteins 0.000 description 1
- 102000004020 Oxygenases Human genes 0.000 description 1
- 241000228143 Penicillium Species 0.000 description 1
- 108091005804 Peptidases Proteins 0.000 description 1
- 102000035195 Peptidases Human genes 0.000 description 1
- 102000003992 Peroxidases Human genes 0.000 description 1
- 108090000472 Phosphoenolpyruvate carboxykinase (ATP) Proteins 0.000 description 1
- 102000011025 Phosphoglycerate Mutase Human genes 0.000 description 1
- 108090000608 Phosphoric Monoester Hydrolases Proteins 0.000 description 1
- 102000004160 Phosphoric Monoester Hydrolases Human genes 0.000 description 1
- 241000235648 Pichia Species 0.000 description 1
- 102000013566 Plasminogen Human genes 0.000 description 1
- 108010051456 Plasminogen Proteins 0.000 description 1
- 102000001938 Plasminogen Activators Human genes 0.000 description 1
- 108010001014 Plasminogen Activators Proteins 0.000 description 1
- ZLMJMSJWJFRBEC-UHFFFAOYSA-N Potassium Chemical compound [K] ZLMJMSJWJFRBEC-UHFFFAOYSA-N 0.000 description 1
- GOOHAUXETOMSMM-UHFFFAOYSA-N Propylene oxide Chemical compound CC1CO1 GOOHAUXETOMSMM-UHFFFAOYSA-N 0.000 description 1
- 241001528479 Pseudoflavonifractor capillosus Species 0.000 description 1
- 241000589517 Pseudomonas aeruginosa Species 0.000 description 1
- 101000937808 Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) Beta-alanine-pyruvate aminotransferase Proteins 0.000 description 1
- 101000593541 Pseudomonas putida Omega-amino acid-pyruvate aminotransferase Proteins 0.000 description 1
- 241000736843 Pyrobaculum aerophilum Species 0.000 description 1
- 241000205160 Pyrococcus Species 0.000 description 1
- 102000004879 Racemases and epimerases Human genes 0.000 description 1
- 108090001066 Racemases and epimerases Proteins 0.000 description 1
- 241000700157 Rattus norvegicus Species 0.000 description 1
- 102000007056 Recombinant Fusion Proteins Human genes 0.000 description 1
- 108010008281 Recombinant Fusion Proteins Proteins 0.000 description 1
- 238000005602 Reppe reaction Methods 0.000 description 1
- 241001148115 Rhizobium etli Species 0.000 description 1
- 241000589194 Rhizobium leguminosarum Species 0.000 description 1
- 240000005384 Rhizopus oryzae Species 0.000 description 1
- 235000013752 Rhizopus oryzae Nutrition 0.000 description 1
- 241000191025 Rhodobacter Species 0.000 description 1
- 241000711837 Roseburia sp. Species 0.000 description 1
- 241000516658 Roseiflexus castenholzii Species 0.000 description 1
- DFPOZTRSOAQFIK-UHFFFAOYSA-N S,S-dimethyl-beta-propiothetin Chemical compound C[S+](C)CCC([O-])=O DFPOZTRSOAQFIK-UHFFFAOYSA-N 0.000 description 1
- 244000253911 Saccharomyces fragilis Species 0.000 description 1
- 235000018368 Saccharomyces fragilis Nutrition 0.000 description 1
- 241000293869 Salmonella enterica subsp. enterica serovar Typhimurium Species 0.000 description 1
- 241000605031 Selenomonas ruminantium Species 0.000 description 1
- 238000012300 Sequence Analysis Methods 0.000 description 1
- MTCFGRXMJLQNBG-UHFFFAOYSA-N Serine Natural products OCC(N)C(O)=O MTCFGRXMJLQNBG-UHFFFAOYSA-N 0.000 description 1
- 241000607715 Serratia marcescens Species 0.000 description 1
- 241000221095 Simmondsia Species 0.000 description 1
- 241000194020 Streptococcus thermophilus Species 0.000 description 1
- 241000187747 Streptomyces Species 0.000 description 1
- 241000187432 Streptomyces coelicolor Species 0.000 description 1
- 241000187392 Streptomyces griseus Species 0.000 description 1
- 108050003834 Succinate CoA transferases Proteins 0.000 description 1
- 102000019259 Succinate Dehydrogenase Human genes 0.000 description 1
- 108010012901 Succinate Dehydrogenase Proteins 0.000 description 1
- 241000205098 Sulfolobus acidocaldarius Species 0.000 description 1
- 241000205091 Sulfolobus solfataricus Species 0.000 description 1
- 241000160715 Sulfolobus tokodaii Species 0.000 description 1
- 241000282898 Sus scrofa Species 0.000 description 1
- 208000003217 Tetany Diseases 0.000 description 1
- 241001147775 Thermoanaerobacter brockii Species 0.000 description 1
- 241000204666 Thermotoga maritima Species 0.000 description 1
- 102000003978 Tissue Plasminogen Activator Human genes 0.000 description 1
- 108090000373 Tissue Plasminogen Activator Proteins 0.000 description 1
- HDTRYLNUVZCQOY-WSWWMNSNSA-N Trehalose Natural products O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@@H]1O[C@@H]1[C@H](O)[C@@H](O)[C@@H](O)[C@@H](CO)O1 HDTRYLNUVZCQOY-WSWWMNSNSA-N 0.000 description 1
- 241000589892 Treponema denticola Species 0.000 description 1
- 241000224526 Trichomonas Species 0.000 description 1
- 241000224527 Trichomonas vaginalis Species 0.000 description 1
- 102000005924 Triose-Phosphate Isomerase Human genes 0.000 description 1
- 108700015934 Triose-phosphate isomerases Proteins 0.000 description 1
- 241000223109 Trypanosoma cruzi Species 0.000 description 1
- 241001034637 Tsukamurella paurometabola DSM 20162 Species 0.000 description 1
- 102100021869 Tyrosine aminotransferase Human genes 0.000 description 1
- 102000016540 Tyrosine aminotransferases Human genes 0.000 description 1
- 108050006053 Tyrosine aminotransferases Proteins 0.000 description 1
- 108010042606 Tyrosine transaminase Proteins 0.000 description 1
- 241000607626 Vibrio cholerae Species 0.000 description 1
- 241000607272 Vibrio parahaemolyticus Species 0.000 description 1
- 241000607265 Vibrio vulnificus Species 0.000 description 1
- 241000405217 Viola <butterfly> Species 0.000 description 1
- 241000289690 Xenarthra Species 0.000 description 1
- 241000588901 Zymomonas Species 0.000 description 1
- 241000029538 [Mannheimia] succiniciproducens Species 0.000 description 1
- XJLXINKUBYWONI-DQQFMEOOSA-N [[(2r,3r,4r,5r)-5-(6-aminopurin-9-yl)-3-hydroxy-4-phosphonooxyoxolan-2-yl]methoxy-hydroxyphosphoryl] [(2s,3r,4s,5s)-5-(3-carbamoylpyridin-1-ium-1-yl)-3,4-dihydroxyoxolan-2-yl]methyl phosphate Chemical compound NC(=O)C1=CC=C[N+]([C@@H]2[C@H]([C@@H](O)[C@H](COP([O-])(=O)OP(O)(=O)OC[C@@H]3[C@H]([C@@H](OP(O)(O)=O)[C@@H](O3)N3C4=NC=NC(N)=C4N=C3)O)O2)O)=C1 XJLXINKUBYWONI-DQQFMEOOSA-N 0.000 description 1
- 101150077561 aceF gene Proteins 0.000 description 1
- 230000021736 acetylation Effects 0.000 description 1
- 238000006640 acetylation reaction Methods 0.000 description 1
- 230000009471 action Effects 0.000 description 1
- 239000000654 additive Substances 0.000 description 1
- 101150014383 adhE gene Proteins 0.000 description 1
- 239000008272 agar Substances 0.000 description 1
- 230000001476 alcoholic effect Effects 0.000 description 1
- PPQRONHOSHZGFQ-LMVFSUKVSA-N aldehydo-D-ribose 5-phosphate Chemical compound OP(=O)(O)OC[C@@H](O)[C@@H](O)[C@@H](O)C=O PPQRONHOSHZGFQ-LMVFSUKVSA-N 0.000 description 1
- PNNNRSAQSRJVSB-BXKVDMCESA-N aldehydo-L-rhamnose Chemical compound C[C@H](O)[C@H](O)[C@@H](O)[C@@H](O)C=O PNNNRSAQSRJVSB-BXKVDMCESA-N 0.000 description 1
- PYMYPHUHKUWMLA-WISUUJSJSA-N aldehydo-L-xylose Chemical compound OC[C@H](O)[C@@H](O)[C@H](O)C=O PYMYPHUHKUWMLA-WISUUJSJSA-N 0.000 description 1
- 229930195726 aldehydo-L-xylose Natural products 0.000 description 1
- 150000001323 aldoses Chemical class 0.000 description 1
- 239000003513 alkali Substances 0.000 description 1
- 150000001336 alkenes Chemical class 0.000 description 1
- HDTRYLNUVZCQOY-LIZSDCNHSA-N alpha,alpha-trehalose Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)O[C@@H]1O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 HDTRYLNUVZCQOY-LIZSDCNHSA-N 0.000 description 1
- 102000012086 alpha-L-Fucosidase Human genes 0.000 description 1
- 108010061314 alpha-L-Fucosidase Proteins 0.000 description 1
- OBETXYAYXDNJHR-UHFFFAOYSA-N alpha-ethylcaproic acid Natural products CCCCC(CC)C(O)=O OBETXYAYXDNJHR-UHFFFAOYSA-N 0.000 description 1
- 230000004075 alteration Effects 0.000 description 1
- 238000005576 amination reaction Methods 0.000 description 1
- 125000003277 amino group Chemical group 0.000 description 1
- 229940067621 aminobutyrate Drugs 0.000 description 1
- AVKUERGKIZMTKX-NJBDSQKTSA-N ampicillin Chemical compound C1([C@@H](N)C(=O)N[C@H]2[C@H]3SC([C@@H](N3C2=O)C(O)=O)(C)C)=CC=CC=C1 AVKUERGKIZMTKX-NJBDSQKTSA-N 0.000 description 1
- 230000009604 anaerobic growth Effects 0.000 description 1
- 239000003242 anti bacterial agent Substances 0.000 description 1
- 229940088710 antibiotic agent Drugs 0.000 description 1
- ODKSFYDXXFIFQN-UHFFFAOYSA-N arginine Natural products OC(=O)C(N)CCCNC(N)=N ODKSFYDXXFIFQN-UHFFFAOYSA-N 0.000 description 1
- 235000009697 arginine Nutrition 0.000 description 1
- 101150010999 aroP gene Proteins 0.000 description 1
- 150000001491 aromatic compounds Chemical class 0.000 description 1
- 210000004507 artificial chromosome Anatomy 0.000 description 1
- 125000003118 aryl group Chemical group 0.000 description 1
- 238000003556 assay Methods 0.000 description 1
- 101150063145 atoA gene Proteins 0.000 description 1
- 101150008413 atoD gene Proteins 0.000 description 1
- GONOPSZTUGRENK-UHFFFAOYSA-N benzyl(trichloro)silane Chemical compound Cl[Si](Cl)(Cl)CC1=CC=CC=C1 GONOPSZTUGRENK-UHFFFAOYSA-N 0.000 description 1
- BGWGXPAPYGQALX-ARQDHWQXSA-N beta-D-fructofuranose 6-phosphate Chemical compound OC[C@@]1(O)O[C@H](COP(O)(O)=O)[C@@H](O)[C@@H]1O BGWGXPAPYGQALX-ARQDHWQXSA-N 0.000 description 1
- RTGHRDFWYQHVFW-UHFFFAOYSA-N beta-Ketoadipic acid Natural products OC(=O)CCC(=O)CC(O)=O RTGHRDFWYQHVFW-UHFFFAOYSA-N 0.000 description 1
- 230000008827 biological function Effects 0.000 description 1
- 230000008236 biological pathway Effects 0.000 description 1
- 230000033228 biological regulation Effects 0.000 description 1
- 238000010504 bond cleavage reaction Methods 0.000 description 1
- 239000001273 butane Substances 0.000 description 1
- CRFNGMNYKDXRTN-CITAKDKDSA-N butyryl-CoA Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCSC(=O)CCC)O[C@H]1N1C2=NC=NC(N)=C2N=C1 CRFNGMNYKDXRTN-CITAKDKDSA-N 0.000 description 1
- TWFZGCMQGLPBSX-UHFFFAOYSA-N carbendazim Chemical compound C1=CC=C2NC(NC(=O)OC)=NC2=C1 TWFZGCMQGLPBSX-UHFFFAOYSA-N 0.000 description 1
- 150000001721 carbon Chemical group 0.000 description 1
- 150000001722 carbon compounds Chemical class 0.000 description 1
- 239000001569 carbon dioxide Substances 0.000 description 1
- 229910002092 carbon dioxide Inorganic materials 0.000 description 1
- 239000011203 carbon fibre reinforced carbon Substances 0.000 description 1
- 229910002091 carbon monoxide Inorganic materials 0.000 description 1
- 150000001728 carbonyl compounds Chemical class 0.000 description 1
- 150000001733 carboxylic acid esters Chemical class 0.000 description 1
- 230000006652 catabolic pathway Effects 0.000 description 1
- 238000010531 catalytic reduction reaction Methods 0.000 description 1
- MLYYVTUWGNIJIB-BXKDBHETSA-N cefazolin Chemical compound S1C(C)=NN=C1SCC1=C(C(O)=O)N2C(=O)[C@@H](NC(=O)CN3N=NN=C3)[C@H]2SC1 MLYYVTUWGNIJIB-BXKDBHETSA-N 0.000 description 1
- 238000004113 cell culture Methods 0.000 description 1
- 230000010261 cell growth Effects 0.000 description 1
- 230000004098 cellular respiration Effects 0.000 description 1
- 238000012512 characterization method Methods 0.000 description 1
- 239000012707 chemical precursor Substances 0.000 description 1
- 229960005091 chloramphenicol Drugs 0.000 description 1
- WIIZWVCIJKGZOK-RKDXNWHRSA-N chloramphenicol Chemical compound ClC(Cl)C(=O)N[C@H](CO)[C@H](O)C1=CC=C([N+]([O-])=O)C=C1 WIIZWVCIJKGZOK-RKDXNWHRSA-N 0.000 description 1
- OEYIOHPDSNJKLS-UHFFFAOYSA-N choline Chemical compound C[N+](C)(C)CCO OEYIOHPDSNJKLS-UHFFFAOYSA-N 0.000 description 1
- 229960001231 choline Drugs 0.000 description 1
- 238000010367 cloning Methods 0.000 description 1
- 230000004186 co-expression Effects 0.000 description 1
- 238000009833 condensation Methods 0.000 description 1
- 230000005494 condensation Effects 0.000 description 1
- 230000021615 conjugation Effects 0.000 description 1
- 208000012839 conversion disease Diseases 0.000 description 1
- 229920001577 copolymer Polymers 0.000 description 1
- LDHQCZJRKDOVOX-NSCUHMNNSA-N crotonic acid Chemical compound C\C=C\C(O)=O LDHQCZJRKDOVOX-NSCUHMNNSA-N 0.000 description 1
- XUJNEKJLAYXESH-UHFFFAOYSA-N cysteine Natural products SCC(N)C(O)=O XUJNEKJLAYXESH-UHFFFAOYSA-N 0.000 description 1
- 235000018417 cysteine Nutrition 0.000 description 1
- 210000000172 cytosol Anatomy 0.000 description 1
- 229940022769 d- lactic acid Drugs 0.000 description 1
- 238000006392 deoxygenation reaction Methods 0.000 description 1
- 238000006477 desulfuration reaction Methods 0.000 description 1
- 230000023556 desulfurization Effects 0.000 description 1
- 238000001514 detection method Methods 0.000 description 1
- 150000004985 diamines Chemical class 0.000 description 1
- RAYJUFCFJUVJBB-UHFFFAOYSA-N dihydrokaempferol Natural products OC1Oc2c(O)cc(O)cc2C(=O)C1c3ccc(O)cc3 RAYJUFCFJUVJBB-UHFFFAOYSA-N 0.000 description 1
- 125000000118 dimethyl group Chemical group [H]C([H])([H])* 0.000 description 1
- BEPAFCGSDWSTEL-UHFFFAOYSA-N dimethyl malonate Chemical compound COC(=O)CC(=O)OC BEPAFCGSDWSTEL-UHFFFAOYSA-N 0.000 description 1
- WQXNXVUDBPYKBA-YFKPBYRVSA-N ectoine Chemical compound CC1=[NH+][C@H](C([O-])=O)CCN1 WQXNXVUDBPYKBA-YFKPBYRVSA-N 0.000 description 1
- 239000012636 effector Substances 0.000 description 1
- 238000004520 electroporation Methods 0.000 description 1
- 239000003623 enhancer Substances 0.000 description 1
- 230000002708 enhancing effect Effects 0.000 description 1
- 229940092559 enterobacter aerogenes Drugs 0.000 description 1
- 230000032050 esterification Effects 0.000 description 1
- 238000005886 esterification reaction Methods 0.000 description 1
- 150000002148 esters Chemical class 0.000 description 1
- UKFXDFUAPNAMPJ-UHFFFAOYSA-N ethylmalonic acid Chemical compound CCC(C(O)=O)C(O)=O UKFXDFUAPNAMPJ-UHFFFAOYSA-N 0.000 description 1
- 230000001747 exhibiting effect Effects 0.000 description 1
- 230000004129 fatty acid metabolism Effects 0.000 description 1
- 230000004136 fatty acid synthesis Effects 0.000 description 1
- 230000002349 favourable effect Effects 0.000 description 1
- 230000002538 fungal effect Effects 0.000 description 1
- 239000007789 gas Substances 0.000 description 1
- 238000004817 gas chromatography Methods 0.000 description 1
- 238000010353 genetic engineering Methods 0.000 description 1
- 235000003869 genetically modified organism Nutrition 0.000 description 1
- 238000011331 genomic analysis Methods 0.000 description 1
- 239000006481 glucose medium Substances 0.000 description 1
- 230000004190 glucose uptake Effects 0.000 description 1
- WHUUTDBJXJRKMK-VKHMYHEASA-L glutamate group Chemical group N[C@@H](CCC(=O)[O-])C(=O)[O-] WHUUTDBJXJRKMK-VKHMYHEASA-L 0.000 description 1
- 150000002337 glycosamines Chemical class 0.000 description 1
- 125000000623 heterocyclic group Chemical group 0.000 description 1
- 108090001018 hexadecanal dehydrogenase (acylating) Proteins 0.000 description 1
- HNDVDQJCIGZPNO-UHFFFAOYSA-N histidine Natural products OC(=O)C(N)CC1=CN=CN1 HNDVDQJCIGZPNO-UHFFFAOYSA-N 0.000 description 1
- 235000014304 histidine Nutrition 0.000 description 1
- 229930195733 hydrocarbon Natural products 0.000 description 1
- 238000007327 hydrogenolysis reaction Methods 0.000 description 1
- 230000007062 hydrolysis Effects 0.000 description 1
- 238000006460 hydrolysis reaction Methods 0.000 description 1
- 125000002887 hydroxy group Chemical group [H]O* 0.000 description 1
- 125000004029 hydroxymethyl group Chemical group [H]OC([H])([H])* 0.000 description 1
- 238000003119 immunoblot Methods 0.000 description 1
- 238000001727 in vivo Methods 0.000 description 1
- 230000002779 inactivation Effects 0.000 description 1
- 239000004434 industrial solvent Substances 0.000 description 1
- ODBLHEXUDAPZAU-UHFFFAOYSA-N isocitric acid Chemical compound OC(=O)C(O)C(C(O)=O)CC(O)=O ODBLHEXUDAPZAU-UHFFFAOYSA-N 0.000 description 1
- 229960000310 isoleucine Drugs 0.000 description 1
- AGPKZVBTJJNPAG-UHFFFAOYSA-N isoleucine Natural products CCC(C)C(N)C(O)=O AGPKZVBTJJNPAG-UHFFFAOYSA-N 0.000 description 1
- 150000004723 keto acid derivatives Chemical class 0.000 description 1
- 150000002576 ketones Chemical class 0.000 description 1
- 150000002584 ketoses Chemical class 0.000 description 1
- 235000020061 kirsch Nutrition 0.000 description 1
- 229940031154 kluyveromyces marxianus Drugs 0.000 description 1
- HXEACLLIILLPRG-RXMQYKEDSA-N l-pipecolic acid Natural products OC(=O)[C@H]1CCCCN1 HXEACLLIILLPRG-RXMQYKEDSA-N 0.000 description 1
- 229940072205 lactobacillus plantarum Drugs 0.000 description 1
- 150000002596 lactones Chemical class 0.000 description 1
- 229920005610 lignin Polymers 0.000 description 1
- 238000009630 liquid culture Methods 0.000 description 1
- 229940049920 malate Drugs 0.000 description 1
- 108010089734 malonyl-CoA synthetase Proteins 0.000 description 1
- 238000001819 mass spectrum Methods 0.000 description 1
- 239000000463 material Substances 0.000 description 1
- 239000011159 matrix material Substances 0.000 description 1
- 230000007246 mechanism Effects 0.000 description 1
- 230000001404 mediated effect Effects 0.000 description 1
- 108020004999 messenger RNA Proteins 0.000 description 1
- 230000004066 metabolic change Effects 0.000 description 1
- 238000001466 metabolic labeling Methods 0.000 description 1
- 239000000203 mixture Substances 0.000 description 1
- 238000010369 molecular cloning Methods 0.000 description 1
- 238000000302 molecular modelling Methods 0.000 description 1
- IJDNQMDRQITEOD-UHFFFAOYSA-N n-butane Chemical compound CCCC IJDNQMDRQITEOD-UHFFFAOYSA-N 0.000 description 1
- FUZZWVXGSFPDMH-UHFFFAOYSA-N n-hexanoic acid Natural products CCCCCC(O)=O FUZZWVXGSFPDMH-UHFFFAOYSA-N 0.000 description 1
- OFBQJSOFQDEBGM-UHFFFAOYSA-N n-pentane Natural products CCCCC OFBQJSOFQDEBGM-UHFFFAOYSA-N 0.000 description 1
- 210000003463 organelle Anatomy 0.000 description 1
- 230000000065 osmolyte Effects 0.000 description 1
- 230000001590 oxidative effect Effects 0.000 description 1
- 150000002924 oxiranes Chemical class 0.000 description 1
- 108040007629 peroxidase activity proteins Proteins 0.000 description 1
- 239000003208 petroleum Substances 0.000 description 1
- 239000003279 phenylacetic acid Substances 0.000 description 1
- 229960003424 phenylacetic acid Drugs 0.000 description 1
- 108010025593 phenylalanine (histidine) aminotransferase Proteins 0.000 description 1
- NBIIXXVUZAFLBC-UHFFFAOYSA-K phosphate Chemical compound [O-]P([O-])([O-])=O NBIIXXVUZAFLBC-UHFFFAOYSA-K 0.000 description 1
- 239000008363 phosphate buffer Substances 0.000 description 1
- 229940127126 plasminogen activator Drugs 0.000 description 1
- 150000003071 polychlorinated biphenyls Chemical class 0.000 description 1
- 239000011591 potassium Substances 0.000 description 1
- 229910052700 potassium Inorganic materials 0.000 description 1
- 125000002924 primary amino group Chemical group [H]N([H])* 0.000 description 1
- 235000019833 protease Nutrition 0.000 description 1
- 239000003223 protective agent Substances 0.000 description 1
- HNJBEVLQSNELDL-UHFFFAOYSA-N pyrrolidin-2-one Chemical compound O=C1CCCN1 HNJBEVLQSNELDL-UHFFFAOYSA-N 0.000 description 1
- 150000004728 pyruvic acid derivatives Chemical class 0.000 description 1
- 239000002994 raw material Substances 0.000 description 1
- 238000005215 recombination Methods 0.000 description 1
- 230000010076 replication Effects 0.000 description 1
- 238000011160 research Methods 0.000 description 1
- 229920005989 resin Polymers 0.000 description 1
- 239000011347 resin Substances 0.000 description 1
- 230000029058 respiratory gaseous exchange Effects 0.000 description 1
- 230000000284 resting effect Effects 0.000 description 1
- 238000007363 ring formation reaction Methods 0.000 description 1
- 102200120552 rs121912529 Human genes 0.000 description 1
- 238000007789 sealing Methods 0.000 description 1
- 235000004400 serine Nutrition 0.000 description 1
- 150000004666 short chain fatty acids Chemical class 0.000 description 1
- 235000021391 short chain fatty acids Nutrition 0.000 description 1
- 238000004088 simulation Methods 0.000 description 1
- 239000011734 sodium Substances 0.000 description 1
- 229910052708 sodium Inorganic materials 0.000 description 1
- 159000000000 sodium salts Chemical class 0.000 description 1
- 244000000000 soil microbiome Species 0.000 description 1
- 238000007619 statistical method Methods 0.000 description 1
- 108010030426 succinyl-CoA hydrolase Proteins 0.000 description 1
- 150000008163 sugars Chemical class 0.000 description 1
- 239000006228 supernatant Substances 0.000 description 1
- 150000007970 thio esters Chemical class 0.000 description 1
- 229960000187 tissue plasminogen activator Drugs 0.000 description 1
- 239000003053 toxin Substances 0.000 description 1
- 231100000765 toxin Toxicity 0.000 description 1
- 108700012359 toxins Proteins 0.000 description 1
- LDHQCZJRKDOVOX-UHFFFAOYSA-N trans-crotonic acid Natural products CC=CC(O)=O LDHQCZJRKDOVOX-UHFFFAOYSA-N 0.000 description 1
- 238000005891 transamination reaction Methods 0.000 description 1
- 238000010361 transduction Methods 0.000 description 1
- 230000026683 transduction Effects 0.000 description 1
- 238000001890 transfection Methods 0.000 description 1
- 238000000844 transformation Methods 0.000 description 1
- 230000001052 transient effect Effects 0.000 description 1
- HGBOYTHUEUWSSQ-UHFFFAOYSA-N valeric aldehyde Natural products CCCCC=O HGBOYTHUEUWSSQ-UHFFFAOYSA-N 0.000 description 1
- 239000013603 viral vector Substances 0.000 description 1
- 238000012070 whole genome sequencing analysis Methods 0.000 description 1
- 239000012130 whole-cell lysate Substances 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/52—Genes encoding for enzymes or proenzymes
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P7/00—Preparation of oxygen-containing organic compounds
- C12P7/02—Preparation of oxygen-containing organic compounds containing a hydroxy group
- C12P7/04—Preparation of oxygen-containing organic compounds containing a hydroxy group acyclic
- C12P7/18—Preparation of oxygen-containing organic compounds containing a hydroxy group acyclic polyhydric
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N1/00—Microorganisms, e.g. protozoa; Compositions thereof; Processes of propagating, maintaining or preserving microorganisms or compositions thereof; Processes of preparing or isolating a composition containing a microorganism; Culture media therefor
- C12N1/20—Bacteria; Culture media therefor
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/0004—Oxidoreductases (1.)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/0004—Oxidoreductases (1.)
- C12N9/0006—Oxidoreductases (1.) acting on CH-OH groups as donors (1.1)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/0004—Oxidoreductases (1.)
- C12N9/001—Oxidoreductases (1.) acting on the CH-CH group of donors (1.3)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/0004—Oxidoreductases (1.)
- C12N9/0012—Oxidoreductases (1.) acting on nitrogen containing compounds as donors (1.4, 1.5, 1.6, 1.7)
- C12N9/0014—Oxidoreductases (1.) acting on nitrogen containing compounds as donors (1.4, 1.5, 1.6, 1.7) acting on the CH-NH2 group of donors (1.4)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/10—Transferases (2.)
- C12N9/1096—Transferases (2.) transferring nitrogenous groups (2.6)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/10—Transferases (2.)
- C12N9/13—Transferases (2.) transferring sulfur containing groups (2.8)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/88—Lyases (4.)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P13/00—Preparation of nitrogen-containing organic compounds
- C12P13/001—Amines; Imines
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P19/00—Preparation of compounds containing saccharide radicals
- C12P19/26—Preparation of nitrogen-containing carbohydrates
- C12P19/28—N-glycosides
- C12P19/38—Nucleosides
- C12P19/40—Nucleosides having a condensed ring system containing a six-membered ring having two nitrogen atoms in the same ring, e.g. purine nucleosides
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P7/00—Preparation of oxygen-containing organic compounds
- C12P7/24—Preparation of oxygen-containing organic compounds containing a carbonyl group
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P7/00—Preparation of oxygen-containing organic compounds
- C12P7/40—Preparation of oxygen-containing organic compounds containing a carboxyl group including Peroxycarboxylic acids
- C12P7/42—Hydroxy-carboxylic acids
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y101/00—Oxidoreductases acting on the CH-OH group of donors (1.1)
- C12Y101/01—Oxidoreductases acting on the CH-OH group of donors (1.1) with NAD+ or NADP+ as acceptor (1.1.1)
- C12Y101/01037—Malate dehydrogenase (1.1.1.37)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y101/00—Oxidoreductases acting on the CH-OH group of donors (1.1)
- C12Y101/01—Oxidoreductases acting on the CH-OH group of donors (1.1) with NAD+ or NADP+ as acceptor (1.1.1)
- C12Y101/01041—Isocitrate dehydrogenase (NAD+) (1.1.1.41)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y101/00—Oxidoreductases acting on the CH-OH group of donors (1.1)
- C12Y101/01—Oxidoreductases acting on the CH-OH group of donors (1.1) with NAD+ or NADP+ as acceptor (1.1.1)
- C12Y101/01042—Isocitrate dehydrogenase (NADP+) (1.1.1.42)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y101/00—Oxidoreductases acting on the CH-OH group of donors (1.1)
- C12Y101/01—Oxidoreductases acting on the CH-OH group of donors (1.1) with NAD+ or NADP+ as acceptor (1.1.1)
- C12Y101/01061—4-Hydroxybutyrate dehydrogenase (1.1.1.61)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y103/00—Oxidoreductases acting on the CH-CH group of donors (1.3)
- C12Y103/01—Oxidoreductases acting on the CH-CH group of donors (1.3) with NAD+ or NADP+ as acceptor (1.3.1)
- C12Y103/01006—Fumarate reductase (NADH) (1.3.1.6)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y401/00—Carbon-carbon lyases (4.1)
- C12Y401/01—Carboxy-lyases (4.1.1)
- C12Y401/01071—2-Oxoglutarate decarboxylase (4.1.1.71)
Landscapes
- Chemical & Material Sciences (AREA)
- Organic Chemistry (AREA)
- Life Sciences & Earth Sciences (AREA)
- Engineering & Computer Science (AREA)
- Wood Science & Technology (AREA)
- Zoology (AREA)
- Health & Medical Sciences (AREA)
- Genetics & Genomics (AREA)
- Bioinformatics & Cheminformatics (AREA)
- General Engineering & Computer Science (AREA)
- Biochemistry (AREA)
- General Health & Medical Sciences (AREA)
- Biotechnology (AREA)
- Microbiology (AREA)
- Chemical Kinetics & Catalysis (AREA)
- General Chemical & Material Sciences (AREA)
- Molecular Biology (AREA)
- Biomedical Technology (AREA)
- Medicinal Chemistry (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Physics & Mathematics (AREA)
- Biophysics (AREA)
- Plant Pathology (AREA)
- Tropical Medicine & Parasitology (AREA)
- Virology (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Preparation Of Compounds By Using Micro-Organisms (AREA)
Abstract
【選択図】図58
Description
本出願は、内容全体が引用により本明細書中に組み込まれている、2009年10月13日に出願された米国特許仮出願第61/251,287号の優先権の利益を主張するものである。
本発明は、1,4-ブタンジオール(BDO)、4-ヒドロキシブタナール(4-HBal)、4-ヒドロキシブチリル-CoA(4-HBCoA)、及び/又はプトレシンを生成するのに十分な量で発現されたBDO、4-HBal及び/又はプトレシン経路酵素をコードしている少なくとも1つの外因性核酸を含むBDO、4-HBal、4-HBCoA及び/又はプトレシン経路を含む非天然微生物体を提供する。この微生物体は、更にBDO、4-HBal、4-HBCoA及び/又はプトレシンの発現を最適化することができる。加えて本発明は、当該微生物体を使用してBDO、4-HBal、4-HBCoA及び/又はプトレシンを生成する方法を提供する。
本発明は、4-ヒドロキシブタン酸(4-HB)、γ-ブチロラクトン、1,4-ブタンジオール(BDO)、4-ヒドロキシブタナール(4-HBal)、4-ヒドロキシブチリル-CoA(4-HBCoA)、及び/又はプトレシンの生合成生成能を有する細胞及び生物体の設計及び生成に関する。本発明は特に、BDO、4-HBal、4-HBCoA及び/又はプトレシン経路酵素をコードしている1つ以上の核酸を導入することによって、BDO、4-HBal、4-HBCoA及び/又はプトレシンを生成することが可能な微生物体の設計に関する。
表1. 4-HBからBDOへの変換のためのアルコールデヒドロゲナーゼ及びアルデヒドデヒドロゲナーゼ
2CO2 + 4H2 + nADP + nPi → CH3COOH + 2H2O + nATP
従って、Wood-Ljungdahl経路を有する非天然微生物は、アセチル-CoA及び他の所望の生成物を生成するためにCO2とH2の混合物をも利用することができる。
(4-ヒドロキシブタン酸の生合成)
本実施例では、4-HB生成のための例示的な生化学合成経路について記載する。
(コハク酸及びアルファ-ケトグルタル酸からの1,4-ブタンジオールの生合成)
本実施例では、微生物体からの4-HB及びBDOの構築及び生合成生成を例示する。4-HB及びBDOのための経路は、本明細書に開示されている。
2Nollingらの文献、J., J. Bacteriol. 183:4823-4838 (2001)
3Pohlmannらの文献、Nat. Biotechnol. 24:1257-1262 (2006)
4Kosakaらの文献、Biosci. Biotechnol. Biochem. 71:58-68 (2007)
5Broschらの文献、Proc. Natl. Acad. Sci. U.S.A. 104:5596-5601 (2007)
6Henneらの文献、Appl. Enviorn. Microbiol. 65:3901-3907 (1999)
pACYC起点及びクロラムフェニコール耐性を有する中コピープラスミドpZA33におけるPlacから発現される遺伝子。
(c)mgタンパク質/ml抽出液として与えられた細胞タンパク質
(4-ヒドロキシブタン酸、γ-ブチロラクトン及び1,4-ブタンジオールの生合成)
本実施例では、発酵及び他のバイオプロセスを使用する4-ヒドロキシブタン酸、γ-ブチロラクトン及び1,4-ブタンジオールの生合成生成について記載する。
(例示的なBDO経路)
本実施例では、1,4-ブタンジオール(BDO)合成経路についての例示的な酵素及び対応する遺伝子を記載する。
アルデヒドからアルコール。アルデヒドからアルコールの変換を触媒する酵素、即ちアルコールデヒドロゲナーゼ又は同等にアルデヒドレダクターゼをコードしている例示的な遺伝子は、C2-C14の中鎖アルコールデヒドロゲナーゼをコードしているalrA(Taniらの文献、Appl. Environ. Microbiol. 66:5231-5235 (2000))、サッカロミセス・セレビシアエ由来のADH2(Atsumiらの文献、Nature 451:86-89 (2008))、C(3)より長い分子を選択するE.コリ由来のyqhD(Sulzenbacherらの文献、Journal of Molecular Biology 342:489-502 (2004))、並びにブチリアルデヒドをブタノールに変換するC.アセトブチリクム由来のbdhI及びbdhII(Walterらの文献、Journal of Bacteriology 174:7149-7158 (1992))を含む。これらの例示的な遺伝子産物の各々についてのタンパク質配列を、入手可能であれば、以下のGenBank寄託番号を使用して見いだすことができる。
アシル-CoAをアルコールに変換する例示的な2工程オキシドレダクターゼは、基質を転換するもの、例えば、アセチル-CoAをエタノールに転換するもの(例えば、E.コリ由来のadhE(Kesskerらの文献、FEBS. Lett. 281:59-63 (1991))、及びブチリル-CoAをブタノールに転換するもの(例えば、C.アセトブチリクム由来のadhE2(Fontaineらの文献、J. Bacteriol. 184:821-830 (2002))を含む。ロイコノストク・メセンテロイデスにおけるadhEによってコードされる酵素は、アセチル-CoAをエタノールに還元することに加えて、分枝鎖化合物イソブチルアルデヒドをイソブチリル-CoAに酸化することが証明された(Kazahayaらの文献、J. Gen.Appl.Microbiol. 18:43-55 (1972);Kooらの文献、Biotechnol Lett. 27:505-510 (2005))。
いくつかのアシル-CoAデヒドロゲナーゼは、アシル-CoAを、その対応するアルデヒドに還元することが可能である。当該酵素をコードしている例示的な遺伝子は、脂肪酸アシル-CoAレダクターゼをコードしているアシネトバクター・カルコアセチカスacr1(Reiser及びSomerville、J. Bacteriology 179:2969-2975 (1997))、アシネトバクター種M-1脂肪酸アシル-CoAレダクターゼ(Ishigeらの文献、Appl. Environ. Microbiol. 68:1192-1195 (2002))、並びにクロストリジウム・クルイベリにおけるsucD遺伝子によってコードされるCoA及びNADP依存性コハク酸セミアルデヒドデヒドロゲナーゼ(Sohling及びGottschalkの文献、J Bacteriol 178:871-80 (1996);Sohling及びGottschalkの文献、J Bacteriol. 178:871-880 (1996))を含む。P.ギンギバリスのSucDは、別のコハク酸セミアルデヒドデヒドロゲナーゼである(Takahashiらの文献、J. Bacteriol. 182:4704-4710 (2000))。bphGによってコード化される、シュードモナス種におけるアセトアルデヒドデヒドロゲナーゼをアシル化する酵素は、アセトアルデヒド、プロピオンアルデヒド、ブチルアルデヒド、イソブチルアルデヒド及びホルムアルデヒドを酸化及びアシル化することが証明されたため、更に別の酵素である(Powlowskiらの文献、J Bacteriol. 175:377-385 (1993))。
この系統の酵素は、1)分枝鎖2-ケト酸デヒドロゲナーゼ、2)アルファ-ケトグルタル酸デヒドロゲナーゼ、及び3)ピルビン酸デヒドロゲナーゼ多酵素複合体(PDHC)を含む。これらの酵素は、2-ケト酸のアシル化酸化的脱炭酸をもたらす一連の部分反応を触媒する多酵素複合体である。2-ケト酸デヒドロゲナーゼ複合体のそれぞれは、中間的代謝における重要な位置を占め、酵素活性は、典型的には厳密に制御される(Friesらの文献、Biochemistry 42:6996-7002 (2003))。それらの酵素は、アルファ-ケト酸デカルボキシラーゼ(E1)、ジヒドロリポアミドアシルトランスフェラーゼ(E2)及びジヒドロリポアミドデヒドロゲナーゼ(E3)の3つの触媒成分の多コピーで構成された複雑であるが、共通の構造体を共有する。E3成分は、生物体における全ての2-ケト酸デヒドロゲナーゼ複合体の間で共有されるが、E1及びE2成分は、異なる遺伝子によってコードされる。酵素成分は、複合体に多くのコピーで存在し、基質チャネリングを介して反応の指向性配列を触媒するために多数の補因子を利用する。これらのデヒドロゲナーゼ複合体の全体的なサイズは非常に大きく、分子質量は、400万〜1000万Daである(即ち、リボソームより大きい)。
このクラスにおける例示的な酵素は、グリセルアルデヒド-3-リン酸をD-グリセレート1,3-ビスリン酸に変換するグリセルアルデヒド3-リン酸デヒドロゲナーゼ(例えば、E.コリgapA(Branlant及びBranlantの文献、Eur. J. Biochem. 150:61-66 (1985)))、L-アスパラギン酸-4-セミアルデヒドをL-4-アスパルチル-リン酸に変換するアスパラギン酸-セミアルデヒドデヒドロゲナーゼ(例えば、E.コリasd(Biellmannらの文献、Eur. J. Biochem. 104:53-58 (1980)))、N-アセチル-L-グルタミン酸-5-セミアルデヒドをN-アセチル-L-グルタミル-5-リン酸に変換するN-アセチル-ガンマ-グルタミル-リン酸レダクターゼ(例えば、E.コリargC(Parsotらの文献、Gene 68:275-283 (1988)))、及びL-グルタミン酸-5-セミアルデヒドをL-グルタミル-5-リン酸に変換するグルタミン酸-5-セミアルデヒドデヒドロゲナーゼ(例えば、E.コリproA(Smithらの文献、J. Bacteriol. 157:545-551 (1984)))を含む。
例示的なエノイル-CoAレダクターゼは、クロトノイル-CoAのブチリル-CoAへの還元を自然に触媒するC.アセトブチリクム由来のbcdの遺伝子産物である(Atsumiらの文献、Metab Eng (2007);Boyntonらの文献、Journal of Bacteriology 178:3015-3024 (1996))。電子伝達フラビンタンパク質をコードしているC.アセトブチリクムetfAB遺伝子の発現と並行してbcdを発現させることによってこの酵素の活性を高めることができる。エノイル-CoAレダクターゼ工程の更なる候補は、E.グラシリス由来のミトコンドリアエノイル-CoAレダクターゼである(Hoffmeisterらの文献、Journal of Biological Chemistry 280:4329-4338 (2005))。そのミトコンドリア標的リーダー配列の除去に続いてこの配列から誘導された構築物をE.コリにクローン化することで活性酵素を得た(Hoffmeisterらの文献、前掲、(2005))。この手法は、真核生物遺伝子、特に、原核生物体において、特定の細胞内区画に遺伝子産物を標的化することができるリーダー配列を有する遺伝子を発現させる技術分野の当業者に周知である。原核生物トレポネマ・デンチコラ由来のこの遺伝子の近いホモログTDE0597は、E.コリにおいてクローン化及び発現された第3のエノイル-CoAレダクターゼを表す(Tucci及びMartinの文献、FEBS Letters 581:1561-1566 (2007))。
アミノ酸に作用するほとんどのオキシドレダクターゼは、NAD+あるいはNADP+を受容体とするアルファ-アミノ酸の酸化的脱アミノ化を触媒する。アミノ酸に作用する例示的なオキシドレダクターゼは、gdhAによってコードされるグルタミン酸デヒドロゲナーゼ(脱アミノ化)、ldhによってコードされるロイシンデヒドロゲナーゼ(脱アミノ化)、及びnadXによってコード化されるアスパラギン酸デヒドロゲナーゼ(脱アミノ化)を含む。エシェリキア・コリ由来のgdhA遺伝子産物(Korberらの文献、J. Mol. Biol. 234:1270-1273 (1993);McPherson及びWoottonの文献、Nucleic. Acids Res. 11:5257-5266 (1983))、サーモトガ・マリティマ由来のgdh(Kortらの文献、Extremophiles 1:52-60 (1997);Lebbinkらの文献、J. Mol. Biol. 280:287-296 (1998);Lebbinkらの文献、J. Mol. Biol. 289:357-369 (1999))、及びハロバクテリウム・サリナルム由来のgdhAl(Ingoldsbyらの文献、Gene 349:237-244 (2005))は、グルタミン酸の2-オキソグルタル酸とアンモニアへの可逆的相互変換を触媒し、それぞれNADP(H)、NAD(H)又はその両方に有利に働く。バシルス・セレウスのldh遺伝子は、ロイシン、イソロイシン、バリン及び2-アミノブタン酸を含む広範な基質を有するLeuDHタンパク質をコードしている(Ansorge及びKulaの文献、 Biotechnol Bioeng. 68:557-562 (2000);Stoyanらの文献、J. Biotechnol 54:77-80 (1997))。アスパラギン酸デヒドロゲナーゼをコードしているサーモトガ・マリティマ由来のnadX遺伝子は、NADの生合成に関与する(Yangらの文献、J.Biol.Chem. 278:8804-8808 (2003))。
例示的なリン酸転移アシルトランスフェラーゼは、ptaによってコードされるホスホトランスアセチラーゼ及びptbによってコードされるホスホトランスブチリラーゼを含む。E.コリ由来のpta遺伝子は、アセチル-CoAをアセチル-リン酸に、又はその逆に変換することができる酵素をコードしている(Suzuki, T.の文献、Biochim. Biophys. Acta 191:559-569(1969))。この酵素は、そのプロセスでアセチル-CoAの代わりにプロピオニル-CoAを利用し、プロピオン酸を形成することもできる(Hesslingerらの文献、Mol. Microbiol 27:477-492 (1998))。同様に、C.アセトブチリクム由来のptb遺伝子は、ブチリル-CoAをブチリル-リン酸に変換することができる酵素をコードしている(Walterらの文献、Gene 134(1):p. 107-11 (1993);Huangらの文献、J Mol Microbiol Biotechnol 2(1):p.33-38 (2000))。更なるptb遺伝子を酪酸産生菌L2-50(Louisらの文献、J. Bacteriol.186:2099-2106 (2004))及びバシルス・メガテリウム(Vazquezらの文献、Curr. Microbiol 42:345-349 (2001))に見いだすことができる。
アスパラギン酸アミノトランスフェラーゼは、アミノ基をアスパラギン酸からアルファ-ケトグルタル酸に転移して、グルタミン酸及びオキサロ酢酸を形成する。この変換は、例えば、エシェリキア・コリ由来のaspC(Yagiらの文献、FEBS Lett. 100:81-84 (1979);Yagiらの文献、Methods Enzymol. 113:83-89 (1985))、サッカロミセス・セレビシアエ由来のAAT2(Yagiらの文献、J Biochem. 92:35-43 (1982))、及びアラビドプシス・サリアナ由来のASP5(48, 108, 225 48. de la et al.、Plant J 46:414-425 (2006);Kwok及びHansonの文献、J Exp. Bot. 55:595-604 (2004);Wilkie及びWarrenの文献、Protein Expr. Purif. 12:381-389 (1998))の遺伝子産物によって触媒される。バリンアミノトランスフェラーゼは、バリン及びピルビン酸の2-ケトイソ吉草酸及びアラニンへの変換を触媒する。E.コリ遺伝子avtAは、1つの当該酵素をコードしている(Whalen及びBergの文献、J. Bacteriol. 150:739-746 (1982))。この遺伝子産物は、また、α-ケト酪酸のアミノ化を触媒し、α-アミノ酪酸を生成するが、この反応におけるアミン供与体は同定されていない(Whalen及びBergの文献、J. Bacteriol. 158:571-574 (1984))。E.コリserCの遺伝子産物は、2つの反応のホスホセリンアミノトランスフェラーゼ及びホスホヒドロキシトレオニンアミノトランスフェラーゼを触媒し(Lam及びWinklerの文献、J. Bacteriol. 172:6518-6528 (1990))、非リン酸化基質に対する活性は検出できなかった(Drewkeらの文献、FEBS. Lett. 390:179-182 (1996))。
例示的なキナーゼは、ackAによってコードされるE.コリ酢酸キナーゼ(Skarstedt及びSilversteinの文献、J. Biol. Chem. 251:6775-6783 (1976))、buk1及びbuk2によってコードされるC.アセトブチリクム酪酸キナーゼ(Walterらの文献、Gene 134(1):107-111 (1993);Huangらの文献、J Mol Microbiol Biotechnol 2(l):33-38 (2000))、及びproBによってコードされるE.コリガンマ-グルタミルキナーゼ(Smithらの文献、J.Bacteriol. 157:545-551 (1984))を含む。これらの酵素は、それぞれ酢酸、酪酸、及びグルタミン酸をリン酸化する。E.コリ由来のackA遺伝子産物は、プロピオン酸をもリン酸化する(Hesslingerらの文献、Mol. Microbiol 27:477-492 (1998))。
CoA-トランスフェラーゼ系統において、酢酸-CoAトランスフェラーゼ(EC2.8.3.8)としても知られるE.コリ酵素アシル-CoA:酢酸-CoAトランスフェラーゼは、イソ酪酸(Matthies及びSchinkの文献、Appl Environ Microbiol 58:1435-1439 (1992))、吉草酸(Vanderwinkelらの文献、Biochem. Biophys. Res Commun. 33:902-908 (1968))、及びブタン酸(Vanderwinkelの文献、前掲(1968))を含む様々な分枝状及び直鎖状アシル-CoA基質からCoA部分を酢酸に転移することが証明された。この酵素は、E.コリ種K12におけるatoA(アルファサブユニット)及びatoD(ベータサブユニット)(Korolevらの文献、Acta Crystallogr.D Biol Crystallogr. 58:2116-2121 (2002);Vanderwinkelの文献、前掲(1968))、並びにコリネバクテリウム・グルタミクムATCC13032におけるactA及びcg0592(Duncanらの文献、Appl Environ Microbiol 68:5186-5190 (2002))によってコードされる。配列相同性によって見いだされる更なる遺伝子は、エシェリキア・コリUT189のatoD及びatoAを含む。
CoAヒドロラーゼ系統において、酵素3-ヒドロキシイソブチリル-CoAヒドロラーゼは、3-HIBCoAに対して特異的であり、バリン分解時に所望の転換を効率的に触媒することが証明された(Shimomuraらの文献、J Biol Chem 269:14248-14253 (1994))。この酵素をコードしている遺伝子は、ラツス・ノルベギクス(Shimomuraらの文献、前掲(1994);Shimomuraらの文献、Methods Enzymol. 324:229-240 (2000))、及びホモサピエンス(Shimomuraらの文献、前掲、2000)のhibchを含む。配列相同性による候補遺伝子は、サッカロミセス・セレビシアエのhibch及びバシルス・セレウスのBC_2292を含む。
例示的なカルボキシ-リアーゼは、2-アセト乳酸をアセトインに変換するクエン酸異化及び分枝鎖アミノ酸生合成に関与するアセト乳酸デカルボキシラーゼである。ラクトコッカス・ラクチスにおいて、該酵素は、遺伝子aldBによってコードされる6つのサブユニットで構成され、バリン、ロイシン及びイソロイシンによって活性化される(Goupilらの文献、Appl. Environ. Microbiol. 62:2636-2640 (1996);Goupil-Feuilleratらの文献、J. Bacteriol. 182:5399-5408 (2000))。この酵素は、E.コリにおいて過剰発現され、特徴づけられた(Phalipらの文献、FEBS Lett. 351:95-99 (1994))。他の生物体において、該酵素は、ストレプトコクス・サーモフィルスのaldC(Monnetらの文献、Lett. Appl. Microbiol. 36:399-405 (2003))、バシルス・ブレビスのaldB(Diderichsenらの文献、J. Bacteriol. 172:4315-4321 (1990);Najmudinらの文献、Acta Crystallogr. D. Biol. Crystallogr. 59:1073-1075 (2003))、及びエンテロバクター・アエロゲネスのbudA(Diderichsenらの文献、J. Bacteriol. 172:4315-4321 (1990))によってコードされる二量体である。バシルス・ブレビス由来の酵素は、バシルス・スブチリスにおいてクローン化及び過剰発現され、結晶学的に特徴づけられた(Najmudinらの文献、Acta Crystallogr. D. Biol. Crystallogr. 59:1073-1075 (2003))。加えて、ロイコノストク・ラクチス由来の酵素は、精製され、特徴づけられたが、遺伝子は単離されていない(O'Sullivanらの文献、FEMS Microbiol. Lett. 194:245-249 (2001))。
ケト酸デカルボキシラーゼとも呼ばれるピルビン酸デカルボキシラーゼ(PDC、EC4.1.1.1)は、ピルビン酸のアセトアルデヒドへの脱炭酸を触媒する、アルコール発酵における重要な酵素である。この酵素は、2-ケト酪酸、2-ケト吉草酸、3-ヒドロキシピルビン酸及び2-フェニルピルビン酸を含む、脂肪族2-ケト酸に対する広い基質範囲を有する(Bergらの文献、Science 318:1782-1786 (2007))。pdcによってコードされるザイモモナス・モビルス由来のPDCは、異なる基質に対する親和性を変化させた指向的操作研究の対象になってきた(Siegertらの文献、Protein Eng Des Sel 18:345-357 (2005))。サッカロミセス・セレビシアエ由来のPDCも広範に研究され、活性の改変のために操作され、E.コリにおいて機能性に発現された((Killenberg-Jabsらの文献、Eur. J. Biochem. 268:1698-1704 (2001);Li及びJordanの文献、Biochemistry 38:10004-10012 (1999);ter Schureらの文献、Appl. Environ. Microbiol. 64:1303-1307 (1998))。この酵素の結晶構造は入手可能である(Killenberg-Jabsの文献、Eur.J.Biochem. 268:1698-1704(2001))。他の十分に特徴づけられたPDC候補は、アセトバクター・パステウリアンス(Chandraらの文献、Arch. Microbiol. 176:443-451 (2001))、及びクルイベロマイセス・ラクチス(Kriegerらの文献、Eur. J. Biochem. 269:3256-3263 (2002))由来の酵素を含む。
ユーバクテリウム・バルケリの2-(ヒドロキシメチル)グルタル酸デヒドラターゼは、例示的なヒドロ-リアーゼである。この酵素は、ニコチン酸異化の観点で研究され、hmdによってコードされる(Alhapelらの文献、Proc Natl Acad Sci USA 103:12341-12346 (2006))。高度な配列相同性を有する類似の酵素が、バクテロイデス・カピロサス、アナエロトルンクス・コリホミニス及びナタラナエロビウス・サーモフィルスに見いだされる。
アスパラギン酸のフマル酸への脱アミノ化を触媒するアスパルターゼ(EC 4.3.1.1)は、微生物において一般的な酵素であり、広く特徴づけられた(Viola, R. E. の文献、Adv. Enzymol. Relat Areas Mol. Biol 74:295-341 (2000))。aspAによってコードされるE.コリアスパルターゼの結晶構造が解明された(Shiらの文献、Biochemistry 36:9136-9144 (1997))。このE.コリ酵素は、代替的な基質のアスパラギン酸フェニルメチルエステル、アスパラギン、アスパラギン酸ベンジル及びリンゴ酸と反応することも示された(Maらの文献、Ann N.Y. Acad Sci 672:60-65 (1992))。個別の研究において、基質特異性を改変させるために、この酵素に対して定方向進化が採用された(Asanoらの文献、Biomol. Eng 22:95-101 (2005))。アスパルターゼ機能性を有する酵素は、ヘモフィルス・インフルエンザ(Sjostromらの文献、Biochim. Biophys. Acta 1324:182-190 (1997))、シュードモナス・フルオレセンス(Takagiらの文献、J. Biochem. 96:545-552 (1984))、バシルス・スブチルス(Sjostromらの文献、Biochim. Biophys. Acta 1324:182-190 (1997))及びセラチア・マルセセンス(Takagi及びKisumiの文献、J Bacteriol. 161:1-6 (1985))においても特徴づけられた。
クロストリジウム・アミノブチリウム及びC.クルイベリの両方に由来する4-ヒドロキシブチリル-CoAデヒドラターゼは、4-ヒドロキシブチリル-CoAのクロトノイル-CoAへの可逆的変換を触媒し、固有のビニルアセチル-CoAΔ-イソメラーゼ活性を保持する(Scherf及びBuckelの文献、Eur. J Biochem. 215:421-429 (1993);Scherfらの文献、Arch. Microbiol 161:239-245 (1994))。N末端アミノ酸配列を含む両原生酵素が精製され、特徴づけられた(Scherf及びBuckelの文献、前掲、1993;Scherfらの文献、前掲、1994)。C.アミノブチリウム及びC.クルイベリ由来のabfD遺伝子は、これらのN末端アミノ酸配列と厳密に一致するため、4-ヒドロキシブチリル-CoAデヒドロゲナーゼ/ビニルアセチル-CoAΔイソメラーゼをコードしている。加えて、ポルフィロモナス・ギンギバリスATCC 33277由来のabfD遺伝子は、ゲノムプロジェクトから相同性を介して同定される。
リジン2,3-アミノムターゼ(EC 5.4.3.2)は、リジンを(3S)-3,6-ジアミノヘキサン酸に変換して、アミン基を2位から3位にシフトさせる例示的なアミノムターゼである。該酵素は、フソバクテリウム・ヌレアツム(kamA)(Barkerらの文献、J. Bacteriol. 152:201-207 (1982))及びクロストリジウム・スブテルミナレ(kamA)(Chirpichらの文献、J. Biol. Chem. 245:1778-1789(1970))を含む、リジンを酢酸及び酪酸に発酵させる細菌に見いだされる。クロストリジウム・スブテルミナレ由来の酵素が結晶化された(Leporeらの文献、Proc. Natl. Acad. Sci.U.S.A 102:13819-13824 (2005))。この機能をコードしている酵素は、バシルス・スブチルスのyodOによってもコードされる(Chenらの文献、Biochem. J. 348 Pt 3:539-549 (2000))。該酵素は、ピリドキサル5'-リン酸を補因子として利用し、S-アデノシルメチオニン(S-Adenosylmethoionine)による活性化を必要とし、L-リジンのみと反応する立体選択性を有する。該酵素は、代替的な基質と反応することが示されていない。
例示的な酸-チオールリガーゼは、インビボで可逆的な反応である、1個のATPのコンカミナント消費を伴うコハク酸からのスクシニル-CoAの形成をともに触媒するE.コリのsucCDの遺伝子産物である(Buckらの文献、Biochemistry 24(22):p.6245-6252 (1985))。更なる例示的なCoA-リガーゼは、配列がまだ特徴づけられていないラットのジカルボン酸-CoAリガーゼ(Vamecqらの文献、Biochem J. 230(3):p. 683-693 (1985))、P.チリソゲヌム由来の2つの特徴づけられたフェニル酢酸-CoAリガーゼ(Lamas-Maceirasらの文献、Biochem J 395(1):147-155 (2006);Wangらの文献、Biochem Biophys Res Commun、360(2):453-458 (2007))、シュードモナス・プチダ由来のフェニル酢酸-CoAリガーゼ(Martinez-Blancoらの文献、J Biol Chem. 265(12):7084-7090 (1990))、及びバシルス・スブチルス由来の6-カルボキシヘキサン酸-CoAリガーゼ(Bowerらの文献、J Bacteriol 178(14):4122-4130 (1996))を含む。
(スクシニル-CoAからの例示的なBDO経路)
本実施例では、スクシニル-CoAからの例示的なBDO経路について記載する。
(アルファ-ケトグルタル酸からの更なる例示的なBDO経路)
本実施例では、アルファ-ケトグルタル酸からの例示的なBDO経路について記載する。
(4-アミノ酪酸からのBDO経路)
本実施例では、4-アミノ酪酸からの例示的なBDO経路について記載する。
(アルファ-ケトグルタル酸の例示的なBDO経路)
本実施例では、アルファ-ケトグルタル酸からの例示的なBDO経路について記載する。
(グルタミン酸からの例示的なBDO経路)
本実施例では、グルタミン酸からの例示的なBDO経路について記載する。
(アセトアセチル-CoAからの例示的なBDO経路)
本実施例では、アセトアセチル-CoAからの例示的なBDO経路について記載する。
(ホモセリンからの例示的なBDO経路)
本実施例では、ホモセリンからの例示的なBDO経路について記載する。
(スクシニル-CoAシンテターゼを発現するBDO産生株)
本実施例では、スクシニル-CoAシンテターゼを発現するBDO産生株におけるBDOの増大された生成について記載する。
(BDO経路酵素をコードしている異種遺伝子の発現)
本実施例では、BDOの生成の向上をもたらす様々な非原生経路酵素の発現について記載する。
(ピルビン酸デヒドロゲナーゼを発現するBDO産生株)
本実施例では、BDO生成を向上させるためのピルビン酸デヒドロゲナーゼ(PDH)の利用について記載する。クレブシエラ・ニューモニエlpdA遺伝子の異種発現を使用してBDO生成を向上させた。
(クエン酸シンターゼ及びアコニターゼを発現するBDO産生株)
本実施例では、クエン酸シンターゼ及びアコニターゼの活性を向上させてBDOの生成を増大させることについて記載する。gltAへのR163L変異は、BDO生成を向上させることが判明した。また、arcAノックアウトを使用してBDO生成を向上させた。
1. 80mol%非標識、20mol%均一-13C
2. 10mol%非標識、90mol%均一-13C
3. 90mol% 1-13C、10mol%均一-13C
4. 40mol% 1-13C、60mol%均一-13C
(BDO菌株発現ホスホエノールピルビン酸カルボキシキナーゼ)
本実施例では、BDO生成を向上させるためのホスホエノールピルビン酸カルボキシキナーゼ(PEPCK)の利用について記載する。ヘモフィルス・インフルエンザPEPCK遺伝子を異種発現に使用した。
(特定の組込み部位におけるBDO経路コード遺伝子の組込み)
本実施例では、より効率的な発現及び安定性を提供するための様々なBDO経路遺伝子のfimD座への組込みについて記載する。
(ピルビン酸副産物の形成を減少するための非ホスホトランスフェラーゼスクロース取込み系の使用)
本実施例では、スクロースのBDOへの変換における副産物としてのピルビン酸を減少させるための非ホスホトランスフェラーゼ(PTS)スクロース取込み系の利用について記載する。
(BDO産生株の概要)
本実施例では、様々なBDO産生株について記載する。
(BDO生成の例示的経路)
本実施例は、BDO経路酵素としてカルボン酸レダクターゼを使用する、4-ヒドロキシブタナール(4-HBal)及び/又はBDOを生成する例示的経路を説明する。
(カルボン酸レダクターゼ酵素を使用する1,4-ブタンジオールの生合成)
本実施例は、カルボン酸レダクターゼ酵素を使用し1,4-ブタンジオールを生成する微生物体の作製を説明する。
表31. 様々な菌株におけるBDO、4-HB及び他の生成物の生成
(カルボン酸レダクターゼを利用するプトレシンへの経路)
本実施例は、カルボン酸レダクターゼを利用する例示的プトレシン経路を説明する。
(C4化合物生成の例示的酵素)
本実施例は、1,4-ブタンジオール、4-ヒドロキシブタナール及びプトレシンなどのC4化合物の生成のための例示的酵素を説明する。
表 32. 図58、62及び63に示された酵素転換のクラス
(アルデヒドからアルコール) 図58、62及び63に記載された3つの転換は、アルデヒドからアルコールへの変換に関与する。これらは、4-ヒドロキシ酪酸デヒドロゲナーゼ(図58及び62の工程C)、1,4-ブタンジオールデヒドロゲナーゼ(図58及び62の工程E)、及び5-ヒドロキシ-2-ペンタン酸(図62の工程Y)である。アルデヒドからアルコールへの変換を触媒する酵素、すなわちアルコールデヒドロゲナーゼ又は同等にアルデヒドレダクターゼをコードしている例示的遺伝子は、C2-C14の中鎖アルコールデヒドロゲナーゼをコードしているalrA(Taniらの文献、Appl. Environ. Microbiol. 66:5231-5235 (2000))、サッカロミセス・セレビシアエ由来のADH2(Atsumiらの文献、Nature 451:86-89 (2008))、C(3)より長い分子に適したE.コリ由来のyqhD(Sulzenbacherらの文献、J. Mol. Biol. 342:489-502 (2004))、及びブチリルアルデヒドをブタノールへ変換するC. アセトブチリクム由来のbdh I及びbdh II(Walterらの文献、J. Bacteriol. 174:7149-7158 (1992))を含む。各例示的遺伝子産物のタンパク質配列は、下記GenBank寄託番号を使用し認めることができる:
図62の工程S及びWは、それぞれ、4-ヒドロキシ酪酸及び1,4-ブタンジオールを形成することができる二機能性レダクターゼ酵素を示している。アシル-CoAをアルコールへ変換する例示的2工程オキシドレダクターゼは、アセチル-CoAなどの基質をエタノールへ転換する酵素(例えば、E.コリ由来のadhE(Kesslerらの文献、FEBS. Lett. 281:59-63 (1991))、及びブチリル-CoAをブタノールへ転換する酵素(例えば、C.アセトブチリクム由来のadhE2(Fontaine らの文献、J.Bacteriol. 184:821-830 (2002))を含む。C.アセトブチリクムadhE2遺伝子は、4-ヒドロキシブチリル-CoAを1,4-ブタンジオールへ変換することが示された(前記参照)。アセチル-CoAのエタノールへの還元に加え、ロイコノストク・メセンテロイデスにおいてadhEによりコードされた酵素は、分枝鎖化合物イソブチルアルデヒドをイソブチリル-CoAに酸化することが示されている(Kazahayaらの文献、J., Gen. Appl. Microbiol. 18:43-55 (1972);Kooらの文献、Biotechnol. Lett. 27:505-510 (2005))。
図58、62及び63の工程Aは、アルデヒド形成スクシニル-CoAレダクターゼによるスクシニル-CoAのコハク酸セミアルデヒドへの変換に関与している。図62の工程Qは、アルデヒド-形成4-ヒドロキシブチリル-CoAレダクターゼによる4-ヒドロキシブチリル-CoAの4-ヒドロキシブタナールへの変換を示している。いくつかのアシル-CoAデヒドロゲナーゼは、アシル-CoAをその対応するアルデヒドへ還元することが可能である。そのような酵素をコードしている例示的遺伝子は、脂肪酸アシル-CoAレダクターゼをコードしているアシネトバクター・カルコアセチカスacr1(Reiser及びSomervilleの文献、J. Bacteriol. 179:2969-2975 (1997))、アシネトバクター種M-1脂肪酸アシル-CoAレダクターゼ(Ishigeらの文献、Appl. Environ. Microbiol. 68:1192-1195 (2002))、及びクロストリジウム・クルイベリのsucD遺伝子によりコードされたCoA-及びNADP-依存性コハク酸セミアルデヒドデヒドロゲナーゼ(Sohling及びGottschalkの文献、J. Bacteriol. 178:871-80 (1996);Sohling及びGottschalkの文献、J. Bacteriol. 178:871-880 (1996))を含む。P.ギンギバリスのsucDは、別のアルデヒド-形成スクシニル-CoAレダクターゼである(Takahashiらの文献、J.Bacteriol. 182:4704-4710 (2000))。bphGによりコードされたシュードモナス種においてアセトアルデヒドデヒドロゲナーゼをアシル化する酵素は、アセトアルデヒド、プロピオンアルデヒド、ブチルアルデヒド、イソブチルアルデヒド及びホルムアルデヒドを酸化及びアシル化することが実証されているので、更に別の酵素である(Powlowskiらの文献、J. Bacteriol. 175:377-385 (1993))。アセチル-CoAのエタノールへの還元に加え、ロイコノストク・メセンテロイデスのadhEによりコードされた酵素は、分枝鎖化合物イソブチルアルデヒドのイソブチリル-CoAへの酸化を示す(Kooらの文献、Biotechnol. Lett. 27:505-510 (2005))。ブチルアルデヒドデヒドロゲナーゼは、クロストリジウム・サッカロペルブチルアセトニクムなどの溶媒生成性(solventogenic)生物体において、ブチリル-CoAをブチルアルデヒドへ変換する、同様の反応を触媒する(Kosakaらの文献、Biosci. Biotechnol. Biochem. 71:58-68 (2007))。
図62の工程AAは、5-ヒドロキシ-2-オキソペンタン酸の4-ヒドロキシブチリル-CoAへの変換を示している。この転換の候補酵素は、1)分枝鎖2-ケト酸デヒドロゲナーゼ、2)アルファ-ケトグルタル酸デヒドロゲナーゼ、及び3)ピルビン酸デヒドロゲナーゼ多酵素複合体(PDHC)を含む。これらの酵素は、2-ケト酸のアシル化酸化的脱炭酸を生じる一連の部分反応を触媒する多酵素複合体である。2-ケト酸デヒドロゲナーゼ複合体の各々は中間代謝の重要な位置を占め、酵素活性は典型的には厳密に調節される(Fries らの文献、Biochemistry 42:6996-7002 (2003))。これらの酵素は複合体を共有するが、共通の構造は、アルファ-ケト酸デカルボキシラーゼ(E1)、ジヒドロリポアミドアシルトランスフェラーゼ(E2)及びジヒドロリポアミドデヒドロゲナーゼ(E3)の3つの触媒構成成分の多コピーを構成する。このE3構成成分は、生物体の全ての2-ケト酸デヒドロゲナーゼ複合体の間で共有されるが、E1及びE2構成成分は、異なる遺伝子によりコードされる。酵素構成成分は、複合体において多くのコピーで存在し、基質チャネリングを介し反応の指定された配列を触媒するために複数の補因子を利用する。これらのデヒドロゲナーゼ複合体の全体のサイズは非常に大きく、分子質量は400万〜1000万Daである(すなわち、リボソームより大きい)。
4-ヒドロキシブチリル-リン酸の4-ヒドロキシブタナールへの変換は、ECクラス1.2.1のオキシドレダクターゼにより触媒され得る。アスパラギン酸セミアルデヒドデヒドロゲナーゼ(ASD、EC 1.2.1.11)は、4-アスパルチルリン酸のアスパラギン酸-4-セミアルデヒドへのNADPH依存性還元を触媒する。ASDは、アミノ酸生合成に関与し、最近抗微生物薬標的として研究されている(Hadfieldらの文献、Biochemistry 40:14475-14483 (2001))。E.コリASD構造が解明され(Hadfieldらの文献、J. Mol. Biol. 289:991-1002 (1999))、該酵素は、代替基質ベータ-3-メチルアスパルチルリン酸を受け入れることが示された(Shamesらの文献、J. Biol. Chem. 259:15331-15339 (1984))。ヘモフィルス・インフルエンザ酵素は、活性部位での基質結合親和性を変更するための酵素工学研究の対象である(Blancoらの文献、Acta Crystallogr. D. Biol. Crystallogr. 60:1388-1395 (2004);Blancoらの文献、Acta Crystallogr. D. Biol. Crystallogr. 60:1808-1815 (2004))。他のASD候補が、マイコバクテリウム・ツベルクローシス(Shafianiらの文献、J. Appl. Microbiol. 98:832-838 (2005))、メタノコッカス・ジャナスキ(Methanococcus jannaschii)(Faehnleらの文献、J. Mol. Biol. 353:1055-1068 (2005))、感染性微生物ビブリオ・コレラ及びヘリコバクター・ピロリ(Mooreらの文献、Protein Expr. Purif. 25:189-194 (2002))において認められる。関連酵素候補は、S.セレビシアエ(Pauwelsらの文献、Eur. J. Biochem. 270:1014-1024 (2003)、B.スブチリス(O'Reilly及びDevineの文献、Microbiology 140 ( Pt 5):1023-1025 (1994))、及び他の生物体において認められた、アセチルグルタミルリン酸をアセチルグルタミン酸-5-セミアルデヒドに天然に還元する酵素である、アセチルグルタミルリン酸レダクターゼ(EC 1.2.1.38)である。
図58、62及び63のいくつかの工程は、酸レダクターゼによる活性化されない酸のアルデヒドへの変換を示している。これらは、4-ヒドロキシ酪酸、コハク酸、アルファ-ケトグルタル酸、及び4-アミノ酪酸の、各々、4-ヒドロキシブタナール、コハク酸セミアルデヒド、2,5-ジオキソペンタン酸、及び4-アミノブタナールへの変換を含む。1つの注目すべきカルボン酸レダクターゼは、ノカルジア・イオウェンシスにおいて認めることができ、これはカルボン酸のそれらの対応するアルデヒドへのマグネシウム、ATP及びNADPH依存性還元を触媒する(Venkitasubramanianらの文献、J. Biol. Chem. 282:478-485 (2007))。この酵素は、car遺伝子によりコードされ、且つクローン化され、E.コリにおいて機能性に発現された(Venkitasubramanianらの文献、J. Biol. Chem. 282:478-485 (2007))。npt遺伝子産物の発現は、転写後修飾を介して、該酵素の活性を向上した。npt遺伝子は、不活性アポ酵素を活性ハロ酵素に変換する特異的ホスホパンテテイントランスフェラーゼ(PPTase)をコードしている。この酵素の天然の基質はバニリン酸であり、該酵素は、広範な芳香族基質及び脂肪族基質を受け入れる(Venkitasubramanianらの文献、「製薬及びバイオ産業における生触媒」、R.N. Patel編集、第15章、425-440頁、CRC Press LLC, ボカラトン、FL、(2006))。
グルタミン酸デヒドロゲナーゼ(図62及び63工程J)、4-アミノ酪酸デヒドロゲナーゼ(図62及び63工程M)、プトレシンデヒドロゲナーゼ(図63工程D)、5-アミノ-2-オキソペンタン酸デヒドロゲナーゼ(図63工程P)、及びオルニチンデヒドロゲナーゼ(図63工程S)は、オキシドレダクターゼのアミノ化により触媒することができる。このECクラスの酵素は、受容体としてNAD+又はNADP+によるアルファ-アミノ酸の酸化的脱アミノ化を触媒し、これらの反応は典型的には可逆性である。アミノ酸を操作する例示的オキシドレダクターゼは、gdhAによりコードされたグルタミン酸デヒドロゲナーゼ(脱アミノ化)、ldhによりコードされたロイシンデヒドロゲナーゼ(脱アミノ化)、及びnadXによりコードされたアスパラギン酸デヒドロゲナーゼ(脱アミノ化)がある。エシェリキア・コリ由来のgdhA遺伝子産物(Korberらの文献、J. Mol. Biol. 234:1270-1273 (1993);McPherson及びWoottonの文献、Nucleic Acids Res. 11:5257-5266 (1983))、サーモトガ・マリティマ由来のgdh(Kortらの文献、Extremophiles 1:52-60 (1997);Lebbinkらの文献、J. Mol. Biol. 280:287-296 (1998);Lebbinkらの文献、J. Mol. Biol. 289:357-369 (1999))、及びハロバクテリウム・サリナルム由来のgdhA1(Ingoldsbyらの文献、Gene 349:237-244 (2005))は、それぞれNADP(H)、NAD(H)、又は両方に嗜好性である一方、グルタミン酸の2-オキソグルタル酸及びアンモニアへの可逆的相互変換を触媒する。バシルス・セレウスのldh遺伝子は、ロイシン、イソロイシン、バリン、及び2-アミノブタン酸を含む広範な基質を有する、LeuDHタンパク質をコードしている(Ansorge及びKulaの文献、Biotechnol. Bioeng. 68:557-562 (2000);Stoyanらの文献、J. Biotechnol. 54:77-80 (1997))。アスパラギン酸デヒドロゲナーゼをコードしているサーモトガ・マリティマ由来のnadX遺伝子は、NADの生合成に関与している(Yangらの文献、J. Biol. Chem. 278:8804-8808 (2003))。
図62の工程Pは、4-ヒドロキシブチリル-CoAの4-ヒドロキシブチリル-Piへの転換を示している。例示的リン酸を転移するアシルトランスフェラーゼは、ptaによりコードされたホスホトランスアセチラーゼ、及びptbによりコードされたホスホトランスブチリラーゼである。E.コリ由来のpta遺伝子は、アセチル-CoAをアセチル-リン酸に、又はその逆に変換することができる酵素をコードしている(Suzukiの文献、Biochim. Biophys. Acta 191:559-569 (1969))。この酵素は、アセチル-CoAの代わりにプロピオニル-CoAを利用することができ、このプロセスにおいてプロピオン酸を形成する(Hesslingerらの文献、Mol. Microbiol. 27:477-492 (1998))。同様に、C.アセトブチリクム由来のptb遺伝子は、ブチリル-CoAをブチリル-リン酸へ変換することができる酵素をコードしている(Walterらの文献、Gene 134:107-111 (1993));Huangらの文献、J Mol. Microbiol. Biotechno.l 2:33-38 (2000))。更なるptb遺伝子は、酪酸産生菌L2-50(Louisらの文献、J. Bacteriol. 186:2099-2106 (2004))、及びバシルス・メガテリウム(Vazquezらの文献、Curr. Microbiol. 42:345-349 (2001))において認めることができる。
アミノトランスフェラーゼは、アルデヒド又はケトンをアミノ基に可逆的に変換する。共通のアミノ供与体/受容体組合せは、グルタミン酸/アルファ-ケトグルタル酸、アラニン/ピルビン酸、及びアスパラギン酸/オキサロ酢酸を含む。いくつかの酵素は、アルデヒドを、4-アミノ酪酸、プトレシン、及び5-アミノ-2-オキソペンタン酸などの第一級アミンへ、又はその逆に変換することが示されている。これらの酵素は、以下の転換を実行するのに特に良く適している:図62及び63の工程N、図63の工程E及びQ。リジン-6-アミノトランスフェラーゼ(EC 2.6.1.36)は、第一級アミンの形成が可能である酵素の一例である。リジンをアルファ-アミノアジピン酸セミアルデヒドに変換するこの酵素機能は、酵母及び細菌において実証されている。カンジダ・ユチリス(Candida utilis)(Hammer及びBodeの文献、J. Basic Microbiol. 32:21-27 (1992))、フラボバクテリウム・ルテセンス(Fujiiらの文献、J. Biochem. 128:391-397 (2000))、及びストレプトマイセス・クラブリゲヌス(Romeroらの文献、J. Ind. Microbiol. Biotechnol. 18:241-246 (1997))由来の候補が特徴付けられている。S.クラブリゲヌス由来の組換えリジン-6-アミノトランスフェラーゼは、E.コリにおいて機能性に発現される(Tobinらの文献、J. Bacteriol. 173:6223-6229 (1991))。F.ルテセンス酵素は、アミノ受容体として、アルファ-ケトグルタル酸に特異的である(Soda及びMisonoの文献、Biochemistry 7:4110-4119 (1968))。アルデヒドを末端アミンへ変換する他の酵素は、2,4-ジアミノブタン酸:2-ケトグルタル酸4-トランスアミナーゼをコードしているアシネトバクター・バウマニのdat遺伝子産物を含む(Ikai及びYamamotoの文献、J. Bacteriol. 179:5118-5125 (1997))。その天然の基質である2,4-ジアミノ酪酸に加え、DATは、リジン、4-アミノ酪酸及びオルニチンの末端アミンをアミノ基転移させる。
ECクラス2.7.2のホスホトランスフェラーゼ酵素は、カルボン酸をホスホン酸に転換すると同時に、1個のATPを加水分解する。図62の工程Oは、そのような酵素による4-ヒドロキシ酪酸の4-ヒドロキシブチリル-リン酸への変換に関与している。酪酸キナーゼ(EC 2.7.2.7)は、C.アセトブチリクムの酸産生時のブチリル-リン酸の酪酸への可逆的変換を実行する(Caryらの文献、Appl. Environ. Microbiol. 56:1576-1583 (1990))。この酵素は、2つのbuk遺伝子産物のいずれかによりコードされている(Huangらの文献、J. Mol. Microbiol. Biotechnol. 2:33-38 (2000))。他の酪酸キナーゼ酵素は、C.ブチリクム及びC. テタノモルフムにおいて認められる(Twarog及びWolfeの文献、J. Bacteriol. 86:112-117 (1963))。サーモトガ・マリティマ由来の関連酵素イソ酪酸キナーゼも、E.コリにおいて発現され、結晶化された(Diaoらの文献、Acta Crystallogr. D. Biol. Crystallogr. 59:1100-1102 (2003);Diao及びHassonの文献、J. Bacteriol. 191:2521-2529 (2009))。アスパルトキナーゼは、アスパラギン酸のATP依存性リン酸化を触媒し、いくつかのアミノ酸の合成に関与している。lysCによりコードされたE.コリのアスパルトキナーゼIII酵素は、広い基質範囲を有し、且つ基質特異性に関与した触媒の残基が解明されている(Keng及びViolaの文献、Arch. Biochem. Biophys. 335:73-81 (1996))。E.コリの2つの追加のキナーゼである酢酸キナーゼ及びガンマ-グルタミルキナーゼも、良い候補である。ackAによりコードされたE.コリ酢酸キナーゼ(Skarstedt及びSilversteinの文献、J. Biol. Chem. 251:6775-6783 (1976))は、酢酸に加えてプロピオン酸をリン酸化する(Hesslingerらの文献、Mol. Microbiol. 27:477-492 (1998))。proBによりコードされたE.コリガンマ-グルタミルキナーゼ(Smithらの文献、J. Bacteriol. 157:545-551 (1984))は、グルタミン酸のガンマ炭酸基(carbonic acid group)をリン酸化する。
クロストリジウム・クルイベリのcat1、cat2、及びcat3の遺伝子産物は、各々、スクシニル-CoA(図62及び63工程G)、4-ヒドロキシブチリル-CoA(図62工程T)、及びブチリル-CoAのアセチルトランスフェラーゼ活性(Seedorfらの文献、Proc. Natl. Acad. Sci. USA 105:2128-2133 (2008);Sohling及びGottschalkの文献、J Bacteriol 178:871-880 (1996))を示すことが記された。同様のCoAトランスフェラーゼ活性は、トリコモナス・バギナリス(van Grinsvenらの文献、J. Biol. Chem. 283:1411-1418 (2008))及びトリパノソーマ・ブルセイ(Riviereらの文献、J. Biol. Chem. 279:45337-45346 (2004))にも存在する。
3.1.2系列の酵素は、アシル-CoA分子をそれらの対応する酸に加水分解する。しかし当該酵素は、プロトンポンプ又は直接ATP加水分解などのエネルギー源に結びつけられる場合、CoA-リガーゼ又はシンテターゼ機能を強調するよう(empart)修飾することができる。いくつかの真核細胞アセチル-CoAヒドロラーゼ(EC 3.1.2.1)は、広い基質特異性を有する。例えばラツス・ノルベギクス脳由来の酵素(Robinsonらの文献、Biochem. Biophys. Res. Commun. 71:959-965 (1976))は、ブチリル-CoA、ヘキサノイル-CoA及びマロニル-CoAと反応する。その配列は報告されていないが、エンドウ葉のミトコンドリア由来の該酵素も広い基質特異性を有し、アセチル-CoA、プロピオニル-CoA、ブチリル-CoA、パルミトイル-CoA、オレオイル-CoA、スクシニル-CoA、及びクロトノイル-CoAに対する活性が実証されている(Zeiher及びRandallの文献、Plant. Physiol. 94:20-27 (1990))。S.セレビシアエ由来のアセチル-CoAヒドロラーゼACH1は、別の候補ヒドロラーゼを表している(Buuらの文献、J. Biol. Chem. 278:17203-17209 (2003))。
(アルファ-ケト酸の脱炭酸) アルファ-ケトグルタル酸デカルボキシラーゼ(図58、62及び63工程B)、5-ヒドロキシ-2-オキソペンタン酸デカルボキシラーゼ(図62工程Z)、及び5-アミノ-2-オキソペンタン酸デカルボキシラーゼ(図63工程R)は全て、アルファ-ケト酸の脱炭酸に関与している。ケト酸の脱炭酸は、ピルビン酸デカルボキシラーゼ(EC 4.1.1.1)、ベンゾイルギ酸デカルボキシラーゼ(EC 4.1.1.7)、アルファ-ケトグルタル酸デカルボキシラーゼ及び分枝鎖アルファ-ケト酸デカルボキシラーゼを含む、変動する基質特異性を持つ様々な酵素により触媒される。
CoAシンテターゼ又はリガーゼの反応は、図62及び63の工程I、並びに図62の工程Vにより必要とされる。コハク酸又は4-ヒドロキシ酪酸は、必要な基質である。恐らくこれらの転換を実行するために酵素をコードしている例示的遺伝子は、スクシニル-CoAシンテターゼ複合体を天然に形成するE.コリのsucCD遺伝子を含む。この酵素複合体は、1個のATPの同時消費によりコハク酸からスクシニル-CoAの形成を天然に触媒し、その反応はインビボにおいて可逆性である(Buckらの文献、Biochem. 24:6245-6252 (1985))。
(カルボン酸レダクターゼを利用するBDO生成)
本実施例は、カルボン酸レダクターゼ酵素を用い1,4-ブタンジオールを生成する微生物体の作製を説明する。
Claims (55)
- 4-ヒドロキシブタナールを生成するのに十分な量で発現される4-ヒドロキシブタナール経路酵素をコードしている外因性核酸を含む4-ヒドロキシブタナール経路を有する微生物体を含む非天然微生物体であって、該4-ヒドロキシブタナール経路が、4-ヒドロキシ酪酸レダクターゼ;スクシニル-CoAレダクターゼ(アルデヒド形成);及び、4-ヒドロキシ酪酸デヒドロゲナーゼを含む、前記非天然微生物体。
- 4-ヒドロキシブタナールを生成するのに十分な量で発現される4-ヒドロキシブタナール経路酵素をコードしている少なくとも1つの外因性核酸を含む4-ヒドロキシブタナール経路を有する微生物体を含む非天然微生物体であって、該4-ヒドロキシブタナール経路が、スクシニル-CoAレダクターゼ(アルデヒド形成);4-ヒドロキシ酪酸デヒドロゲナーゼ;及び、4-ヒドロキシ酪酸レダクターゼを含む、前記非天然微生物体。
- 前記少なくとも1つの外因性核酸が、異種核酸である、請求項1又は2記載の非天然微生物体。
- 請求項1〜3のいずれか1項記載の非天然微生物体を、4-ヒドロキシブタナールを生成するための条件下且つ十分な時間にわたり培養することを含む、4-ヒドロキシブタナールの生成方法。
- 前記非天然微生物体が、実質的に嫌気性培養培地内にある、請求項4記載の方法。
- 4-ヒドロキシブタナールを生成するのに十分な量で発現される4-ヒドロキシブタナール経路酵素をコードしている外因性核酸を含む4-ヒドロキシブタナール経路を有する微生物体を含む非天然微生物体であって、該4-ヒドロキシブタナール経路が、4-ヒドロキシ酪酸レダクターゼ;アルファ-ケトグルタル酸デカルボキシラーゼ;及び、4-ヒドロキシ酪酸デヒドロゲナーゼを含む、前記非天然微生物体。
- 4-ヒドロキシブタナールを生成するのに十分な量で発現される4-ヒドロキシブタナール経路酵素をコードしている少なくとも1つの外因性核酸を含む4-ヒドロキシブタナール経路を有する微生物体を含む非天然微生物体であって、該4-ヒドロキシブタナール経路が、アルファ-ケトグルタル酸デカルボキシラーゼ;4-ヒドロキシ酪酸デヒドロゲナーゼ;及び、4-ヒドロキシ酪酸レダクターゼを含む、前記非天然微生物体。
- 前記少なくとも1つの外因性核酸が、異種核酸である、請求項6又は7のいずれか1項記載の非天然微生物体。
- 請求項6〜8のいずれか1項記載の非天然微生物体を、4-ヒドロキシブタナールを生成するための条件下且つ十分な時間にわたり培養することを含む、4-ヒドロキシブタナールの生成方法。
- 前記非天然微生物体が、実質的に嫌気性培養培地内にある、請求項9記載の方法。
- 4-ヒドロキシブタナールを生成するのに十分な量で発現される4-ヒドロキシブタナール経路酵素をコードしている少なくとも1つの外因性核酸を含む4-ヒドロキシブタナール経路を有する微生物体を含む非天然微生物体であって、該4-ヒドロキシブタナール経路が、コハク酸レダクターゼ;4-ヒドロキシ酪酸デヒドロゲナーゼ;及び、4-ヒドロキシ酪酸レダクターゼを含む、前記非天然微生物体。
- 前記微生物体が、コハク酸レダクターゼ;4-ヒドロキシ酪酸デヒドロゲナーゼ;及び、4-ヒドロキシ酪酸レダクターゼをコードしている3つの外因性核酸を含む、請求項11記載の非天然微生物体。
- 前記少なくとも1つの外因性核酸が、異種核酸である、請求項11記載の非天然微生物体。
- 請求項11〜13のいずれか1項記載の非天然微生物体を、4-ヒドロキシブタナールを生成するための条件下且つ十分な時間にわたり培養することを含む、4-ヒドロキシブタナールの生成方法。
- 前記非天然微生物体が、実質的に嫌気性培養培地内にある、請求項14記載の方法。
- 4-ヒドロキシブタナールを生成するのに十分な量で発現される4-ヒドロキシブタナール経路酵素をコードしている少なくとも1つの外因性核酸を含む4-ヒドロキシブタナール経路を有する微生物体を含む非天然微生物体であって、該4-ヒドロキシブタナール経路が、アルファ-ケトグルタル酸デカルボキシラーゼ、又はグルタミン酸デヒドロゲナーゼ若しくはグルタミン酸トランスアミナーゼ及びグルタミン酸デカルボキシラーゼ及び4-アミノ酪酸デヒドロゲナーゼ又は4-アミノ酪酸トランスアミナーゼ;4-ヒドロキシ酪酸デヒドロゲナーゼ;及び、4-ヒドロキシ酪酸レダクターゼを含む、前記非天然微生物体。
- 前記微生物体が、アルファ-ケトグルタル酸デカルボキシラーゼ、又はグルタミン酸デヒドロゲナーゼ若しくはグルタミン酸トランスアミナーゼ及びグルタミン酸デカルボキシラーゼ及び4-アミノ酪酸デヒドロゲナーゼ又は4-アミノ酪酸トランスアミナーゼ;4-ヒドロキシ酪酸デヒドロゲナーゼ;及び、4-ヒドロキシ酪酸レダクターゼをコードしている少なくとも3つの外因性核酸を含む、請求項16記載の非天然微生物体。
- 前記少なくとも1つの外因性核酸が、異種核酸である、請求項16記載の非天然微生物体。
- 請求項16〜18のいずれか1項記載の非天然微生物体を、4-ヒドロキシブタナールを生成するための条件下且つ十分な時間にわたり培養することを含む、4-ヒドロキシブタナールの生成方法。
- 前記非天然微生物体が、実質的に嫌気性培養培地内にある、請求項19記載の方法。
- 4-ヒドロキシブタナールを生成するのに十分な量で発現される4-ヒドロキシブタナール経路酵素をコードしている少なくとも1つの外因性核酸を含む4-ヒドロキシブタナール経路を有する微生物体を含む非天然微生物体であって、該4-ヒドロキシブタナール経路が、アルファ-ケトグルタル酸レダクターゼ;5-ヒドロキシ-2-オキソペンタン酸デヒドロゲナーゼ;及び、5-ヒドロキシ-2-オキソペンタン酸デカルボキシラーゼを含む、前記非天然微生物体。
- 前記微生物体が、アルファ-ケトグルタル酸レダクターゼ;5-ヒドロキシ-2-オキソペンタン酸デヒドロゲナーゼ;及び、5-ヒドロキシ-2-オキソペンタン酸デカルボキシラーゼをコードしている3つの外因性核酸を含む、請求項21記載の非天然微生物体。
- 前記少なくとも1つの外因性核酸が、異種核酸である、請求項21記載の非天然微生物体。
- 請求項21〜23のいずれか1項記載の非天然微生物体を、4-ヒドロキシブタナールを生成するための条件下且つ十分な時間にわたり培養することを含む、4-ヒドロキシブタナールの生成方法。
- 前記非天然微生物体が、実質的に嫌気性培養培地内にある、請求項24記載の方法。
- プトレシンを生成するのに十分な量で発現されるプトレシン経路酵素をコードしている少なくとも1つの外因性核酸を含むプトレシン経路を有する微生物体を含む非天然微生物体であって、該プトレシン経路が、コハク酸レダクターゼ;4-アミノ酪酸デヒドロゲナーゼ又は4-アミノ酪酸トランスアミナーゼ;4-アミノ酪酸レダクターゼ;及び、プトレシンデヒドロゲナーゼ又はプトレシントランスアミナーゼを含む、前記非天然微生物体。
- 前記微生物体が、コハク酸レダクターゼ;4-アミノ酪酸デヒドロゲナーゼ又は4-アミノ酪酸トランスアミナーゼ;4-アミノ酪酸レダクターゼ;及び、プトレシンデヒドロゲナーゼ又はプトレシントランスアミナーゼをコードしている4つの外因性核酸を含む、請求項26記載の非天然微生物体。
- 前記少なくとも1つの外因性核酸が、異種核酸である、請求項26記載の非天然微生物体。
- 請求項26〜28のいずれか1項記載の非天然微生物体を、プトレシンを生成するための条件下且つ十分な時間にわたり培養することを含む、プトレシンの生成方法。
- 前記非天然微生物体が、実質的に嫌気性培養培地内にある、請求項29記載の方法。
- プトレシンを生成するのに十分な量で発現されるプトレシン経路酵素をコードしている少なくとも1つの外因性核酸を含むプトレシン経路を有する微生物体を含む非天然微生物体であって、該プトレシン経路が、アルファ-ケトグルタル酸デカルボキシラーゼ;4-アミノ酪酸デヒドロゲナーゼ又は4-アミノ酪酸トランスアミナーゼ;4-アミノ酪酸レダクターゼ;及び、プトレシンデヒドロゲナーゼ又はプトレシントランスアミナーゼを含む、前記非天然微生物体。
- 前記微生物体が、アルファ-ケトグルタル酸デカルボキシラーゼ;4-アミノ酪酸デヒドロゲナーゼ又は4-アミノ酪酸トランスアミナーゼ;4-アミノ酪酸レダクターゼ;及び、プトレシンデヒドロゲナーゼ又はプトレシントランスアミナーゼをコードしている4つの外因性核酸を含む、請求項31記載の非天然微生物体。
- 前記少なくとも1つの外因性核酸が、異種核酸である、請求項31記載の非天然微生物体。
- 請求項31〜33のいずれか1項記載の非天然微生物体を、プトレシンを生成するための条件下且つ十分な時間にわたり培養することを含む、プトレシンの生成方法。
- 前記非天然微生物体が、実質的に嫌気性培養培地内にある、請求項34記載の方法。
- プトレシンを生成するのに十分な量で発現されるプトレシン経路酵素をコードしている少なくとも1つの外因性核酸を含むプトレシン経路を有する微生物体を含む非天然微生物体であって、該プトレシン経路が、グルタミン酸デヒドロゲナーゼ又はグルタミン酸トランスアミナーゼ;グルタミン酸デカルボキシラーゼ;4-アミノ酪酸レダクターゼ;及び、プトレシンデヒドロゲナーゼ又はプトレシントランスアミナーゼを含む、前記非天然微生物体。
- 前記微生物体が、グルタミン酸デヒドロゲナーゼ又はグルタミン酸トランスアミナーゼ;グルタミン酸デカルボキシラーゼ;4-アミノ酪酸レダクターゼ;及び、プトレシンデヒドロゲナーゼ又はプトレシントランスアミナーゼをコードしている4つの外因性核酸を含む、請求項36記載の非天然微生物体。
- 前記少なくとも1つの外因性核酸が、異種核酸である、請求項36記載の非天然微生物体。
- 請求項36〜38のいずれか1項記載の非天然微生物体を、プトレシンを生成するための条件下且つ十分な時間にわたり培養することを含む、プトレシンの生成方法。
- 前記非天然微生物体が、実質的に嫌気性培養培地内にある、請求項39記載の方法。
- プトレシンを生成するのに十分な量で発現されるプトレシン経路酵素をコードしている少なくとも1つの外因性核酸を含むプトレシン経路を有する微生物体を含む非天然微生物体であって、該プトレシン経路が、アルファ-ケトグルタル酸レダクターゼ;5-アミノ-2-オキソペンタン酸デヒドロゲナーゼ又は5-アミノ-2-オキソペンタン酸トランスアミナーゼ;5-アミノ-2-オキソペンタン酸デカルボキシラーゼ;及び、プトレシンデヒドロゲナーゼ又はプトレシントランスアミナーゼを含む、前記非天然微生物体。
- 前記微生物体が、アルファ-ケトグルタル酸レダクターゼ;5-アミノ-2-オキソペンタン酸デヒドロゲナーゼ又は5-アミノ-2-オキソペンタン酸トランスアミナーゼ;5-アミノ-2-オキソペンタン酸デカルボキシラーゼ;及び、プトレシンデヒドロゲナーゼ又はプトレシントランスアミナーゼをコードしている4つの外因性核酸を含む、請求項41記載の非天然微生物体。
- 前記少なくとも1つの外因性核酸が、異種核酸である、請求項41記載の非天然微生物体。
- 請求項41〜43のいずれか1項記載の非天然微生物体を、プトレシンを生成するための条件下且つ十分な時間にわたり培養することを含む、プトレシンの生成方法。
- 前記非天然微生物体が、実質的に嫌気性培養培地内にある、請求項44記載の方法。
- プトレシンを生成するのに十分な量で発現されるプトレシン経路酵素をコードしている少なくとも1つの外因性核酸を含むプトレシン経路を有する微生物体を含む非天然微生物体であって、該プトレシン経路が、アルファ-ケトグルタル酸レダクターゼ;5-アミノ-2-オキソペンタン酸デヒドロゲナーゼ又は5-アミノ-2-オキソペンタン酸トランスアミナーゼ;オルニチンデヒドロゲナーゼ又はオルニチントランスアミナーゼ;及び、オルニチンデカルボキシラーゼを含む、前記非天然微生物体。
- 前記微生物体が、アルファ-ケトグルタル酸レダクターゼ;5-アミノ-2-オキソペンタン酸デヒドロゲナーゼ又は5-アミノ-2-オキソペンタン酸トランスアミナーゼ;オルニチンデヒドロゲナーゼ又はオルニチントランスアミナーゼ;及び、オルニチンデカルボキシラーゼをコードしている4つの外因性核酸を含む、請求項46記載の非天然微生物体。
- 前記少なくとも1つの外因性核酸が、異種核酸である、請求項46記載の非天然微生物体。
- 請求項46〜48のいずれか1項記載の非天然微生物体を、プトレシンを生成するための条件下且つ十分な時間にわたり培養することを含む、プトレシンの生成方法。
- 前記非天然微生物体が、実質的に嫌気性培養培地内にある、請求項49記載の方法。
- 4-ヒドロキシブチリル-CoA(4-HBCoA)を生成するのに十分な量で発現される4-ヒドロキシブチリル-CoA経路酵素をコードしている少なくとも1つの外因性核酸を含む4-ヒドロキシブチリル-CoA経路を有する微生物体を含む非天然微生物体であって、該4-ヒドロキシブチリル-CoA経路が、アルファ-ケトグルタル酸レダクターゼ;5-ヒドロキシ-2-オキソペンタン酸デヒドロゲナーゼ;及び、5-ヒドロキシ-2-オキソペンタン酸デヒドロゲナーゼ(脱炭酸)を含む、前記非天然微生物体。
- 前記微生物体が、アルファ-ケトグルタル酸レダクターゼ;5-ヒドロキシ-2-オキソペンタン酸デヒドロゲナーゼ;及び、5-ヒドロキシ-2-オキソペンタン酸デヒドロゲナーゼ(脱炭酸)をコードしている3つの外因性核酸を含む、請求項51記載の非天然微生物体。
- 前記少なくとも1つの外因性核酸が、異種核酸である、請求項51記載の非天然微生物体。
- 請求項51〜53のいずれか1項記載の非天然微生物体を、4-ヒドロキシブチリル-CoAを生成するための条件下且つ十分な時間にわたり培養することを含む、4-ヒドロキシブチリル-CoAの生成方法。
- 前記非天然微生物体が、実質的に嫌気性培養培地内にある、請求項54記載の方法。
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US25128709P | 2009-10-13 | 2009-10-13 | |
| US61/251,287 | 2009-10-13 | ||
| PCT/US2010/052570 WO2011047101A1 (en) | 2009-10-13 | 2010-10-13 | Microorganisms for the production of 1,4-butanediol, 4-hydroxybutanal, 4-hydroxybutyryl-coa, putrescine and related compounds, and methods related thereto |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| JP2013507145A true JP2013507145A (ja) | 2013-03-04 |
| JP2013507145A5 JP2013507145A5 (ja) | 2013-11-28 |
Family
ID=43876524
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2012534339A Pending JP2013507145A (ja) | 2009-10-13 | 2010-10-13 | 1,4−ブタンジオール、4−ヒドロキシブタナール、4−ヒドロキシブチリル−CoA、プトレシン及び関連化合物の生成のための微生物体並びに関連する方法 |
Country Status (10)
| Country | Link |
|---|---|
| US (6) | US8377666B2 (ja) |
| EP (1) | EP2488652A4 (ja) |
| JP (1) | JP2013507145A (ja) |
| KR (1) | KR20120083908A (ja) |
| CN (2) | CN102762735B (ja) |
| AU (1) | AU2010306785B2 (ja) |
| CA (1) | CA2777459A1 (ja) |
| CO (1) | CO6541567A2 (ja) |
| MX (1) | MX2012004291A (ja) |
| WO (1) | WO2011047101A1 (ja) |
Cited By (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| KR20160095053A (ko) * | 2013-12-05 | 2016-08-10 | 알이지 라이프 사이언시스, 엘엘씨 | 지방족 아민의 미생물 생성 |
| JP2021503306A (ja) * | 2017-11-17 | 2021-02-12 | エンザイミカルズ アーゲー | 塩沈殿下のトランスアミナーゼ反応によりカルボニル化合物からアミンを調製する方法 |
Families Citing this family (61)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2009155382A1 (en) | 2008-06-17 | 2009-12-23 | Genomatica, Inc. | Microorganisms and methods for the biosynthesis of fumarate, malate, and acrylate |
| BRPI0915749A2 (pt) * | 2008-07-08 | 2018-07-10 | Opx Biotechnologies Inc | métodos, composições e sistemas para produção biossintética de 1,4-butanodiol |
| JP5912529B2 (ja) | 2008-09-10 | 2016-04-27 | ゲノマチカ, インク. | 1,4−ブタンジオールの生成のための微生物体 |
| KR20110097951A (ko) | 2008-12-16 | 2011-08-31 | 게노마티카 인코포레이티드 | 합성가스와 다른 탄소원을 유용 제품으로 전환시키기 위한 미생물 및 방법 |
| EP3199511B1 (en) | 2009-06-04 | 2020-01-29 | Genomatica, Inc. | Process of separating components of a fermentation broth |
| US8420375B2 (en) * | 2009-06-10 | 2013-04-16 | Genomatica, Inc. | Microorganisms and methods for carbon-efficient biosynthesis of MEK and 2-butanol |
| JP2013507145A (ja) | 2009-10-13 | 2013-03-04 | ゲノマチカ, インク. | 1,4−ブタンジオール、4−ヒドロキシブタナール、4−ヒドロキシブチリル−CoA、プトレシン及び関連化合物の生成のための微生物体並びに関連する方法 |
| CN109136161A (zh) | 2009-12-10 | 2019-01-04 | 基因组股份公司 | 合成气或其他气态碳源和甲醇转化为1,3-丁二醇的方法和有机体 |
| US8445244B2 (en) | 2010-02-23 | 2013-05-21 | Genomatica, Inc. | Methods for increasing product yields |
| AU2012273093A1 (en) * | 2011-06-22 | 2013-05-02 | Genomatica, Inc. | Microorganisms for producing 6-aminocaproic acid |
| AU2012273177A1 (en) | 2011-06-22 | 2013-05-02 | Genomatica, Inc. | Microorganisms for producing 1,3-butanediol and methods related thereto |
| WO2012177601A2 (en) * | 2011-06-22 | 2012-12-27 | Genomatica, Inc. | Microorganisms for producing isobutanol and methods related thereto |
| AU2012272856A1 (en) | 2011-06-22 | 2013-05-02 | Genomatica, Inc. | Microorganisms for producing 1,4-butanediol and methods related thereto |
| KR20140091742A (ko) | 2011-11-11 | 2014-07-22 | 아지노모토 가부시키가이샤 | 발효법에 의한 목적 물질의 제조법 |
| ES2666157T3 (es) | 2012-04-02 | 2018-05-03 | REG Life Sciences, LLC | Enzimas CAR y producción mejorada de alcoholes grasos |
| CN104379734B (zh) | 2012-05-18 | 2018-05-15 | 诺维信公司 | 具有改进的转化效率的细菌突变体 |
| EP2855687B1 (en) * | 2012-06-04 | 2020-04-22 | Genomatica, Inc. | Microorganisms and methods for production of 4-hydroxybutyrate, 1,4-butanediol and related compounds |
| KR102023618B1 (ko) | 2012-07-27 | 2019-09-20 | 삼성전자주식회사 | 1,4-bdo 생성능이 개선된 변이 미생물 및 이를 이용한 1,4-bdo의 제조방법 |
| CN104718282A (zh) | 2012-08-10 | 2015-06-17 | Opx生物工艺学公司 | 用于生产脂肪酸和脂肪酸衍生产物的微生物及方法 |
| US9809833B2 (en) | 2012-09-20 | 2017-11-07 | Bioamber Inc. | Pathways to adipate semialdehyde and other organic products |
| US20150218567A1 (en) | 2012-09-27 | 2015-08-06 | Novozymes A/S | Bacterial Mutants with Improved Transformation Efficiency |
| US11535874B2 (en) * | 2012-10-22 | 2022-12-27 | Genomatica, Inc. | Microorganisms and methods for enhancing the availability of reducing equivalents in the presence of methanol, and for producing succinate related thereto |
| WO2014080683A1 (ja) * | 2012-11-26 | 2014-05-30 | 昭和電工株式会社 | 1,4-ブタンジオールの製造方法及び微生物 |
| CN104797713A (zh) * | 2012-11-27 | 2015-07-22 | 昭和电工株式会社 | 1,4-丁二醇的制造方法及微生物 |
| WO2014087921A1 (ja) * | 2012-12-05 | 2014-06-12 | 昭和電工株式会社 | 1,4-ブタンジオールの製造方法、微生物及び遺伝子 |
| JP6342337B2 (ja) * | 2013-01-21 | 2018-06-13 | 積水化学工業株式会社 | 組換え細胞、並びに、1,4−ブタンジオールの生産方法 |
| KR102349070B1 (ko) * | 2013-03-15 | 2022-01-10 | 카아길, 인코포레이팃드 | 아세틸-CoA 카르복실라아제 돌연변이 |
| WO2014146026A1 (en) | 2013-03-15 | 2014-09-18 | Opx Biotechnologies, Inc. | Bioproduction of chemicals |
| AU2014239256B2 (en) | 2013-03-15 | 2018-11-08 | Genomatica, Inc. | Process and systems for obtaining 1,4-butanediol from fermentation broths |
| WO2014176514A2 (en) * | 2013-04-26 | 2014-10-30 | Genomatica, Inc. | Microorganisms and methods for production of 4-hydroxybutyrate, 1,4-butanediol and related compounds |
| CN103451123B (zh) * | 2013-06-08 | 2015-10-21 | 江苏省农业科学院 | 溶酪大球菌及其制备方法和应用 |
| US11408013B2 (en) | 2013-07-19 | 2022-08-09 | Cargill, Incorporated | Microorganisms and methods for the production of fatty acids and fatty acid derived products |
| CN106795483A (zh) | 2013-07-19 | 2017-05-31 | 嘉吉公司 | 用于生产脂肪酸和脂肪酸衍生产物的微生物及方法 |
| KR101849339B1 (ko) * | 2013-09-30 | 2018-04-17 | 삼성디스플레이 주식회사 | 플렉서블 표시장치 |
| PL3077501T3 (pl) | 2013-12-03 | 2022-01-31 | Genomatica, Inc. | Mikroorganizmy i sposoby poprawy wydajności produktu na metanolu z użyciem syntezy acetylo-coa |
| EP3087174B1 (en) | 2013-12-27 | 2020-05-13 | Genomatica, Inc. | Methods and organisms with increased carbon flux efficiencies |
| US10059920B2 (en) | 2014-01-16 | 2018-08-28 | University Of Delaware | Synthetic methylotrophy to liquid fuels and chemicals |
| US10227616B2 (en) | 2014-04-16 | 2019-03-12 | Novamont S.P.A. | Process for the production of 1,4-butanediol |
| WO2015167043A1 (ko) | 2014-04-30 | 2015-11-05 | 삼성전자 주식회사 | 증가된 알파-케토글루타레이트 데카르복실라제 활성을 갖는 미생물 및 이를 이용한 1,4-부탄디올 생산방법 |
| EP2993228B1 (en) | 2014-09-02 | 2019-10-09 | Cargill, Incorporated | Production of fatty acid esters |
| EP3741865B1 (en) | 2014-09-18 | 2024-03-13 | Genomatica, Inc. | Non-natural microbial organisms with improved energetic efficiency |
| EP3026118A1 (de) * | 2014-11-25 | 2016-06-01 | LANXESS Deutschland GmbH | Enzymatisches Verfahren zur Herstellung von 3-Butenal und 3-Buten-1-ol |
| US10072150B2 (en) * | 2014-12-22 | 2018-09-11 | INVISTA North America S.à r.l. | Methods and materials for the production of monomers for nylon-4/polyester production |
| CA2977593A1 (en) | 2015-02-27 | 2016-09-01 | White Dog Labs, Inc. | Mixotrophic fermentation method for making acetone, isopropanol, butyric acid and other bioproducts, and mixtures thereof |
| CN105441372A (zh) * | 2015-10-30 | 2016-03-30 | 清华大学 | 一种重组谷氨酸棒杆菌与应用 |
| AU2016353242A1 (en) * | 2015-11-12 | 2018-06-21 | Lygos, Inc. | Recombinant host cells and methods for the anaerobic production of L-aspartate and beta-alanine |
| CN106222122A (zh) * | 2016-07-20 | 2016-12-14 | 江南大学 | 大肠杆菌工程菌及其催化马来酸合成富马酸的方法 |
| WO2018144701A2 (en) | 2017-02-02 | 2018-08-09 | Cargill Incorporated | Genetically modified cells that produce c6-c10 fatty acid derivatives |
| BR112019020461A2 (pt) * | 2017-03-31 | 2020-06-09 | Genomatica Inc | variantes de aldeído desidrogenase e métodos de uso |
| US11898172B2 (en) * | 2017-03-31 | 2024-02-13 | Genomatica, Inc. | 3-hydroxybutyryl-CoA dehydrogenase variants and methods of use |
| CN107099558B (zh) * | 2017-04-25 | 2021-06-11 | 华东理工大学 | 一种同时生物合成1,3-丙二醇和乙偶姻的新方法 |
| CN115160545B (zh) | 2017-11-27 | 2024-05-17 | 诺瓦蒙特股份公司 | 用于生产来自可再生资源的1,4-丁二醇的方法和由其获得的聚酯 |
| US20210079334A1 (en) | 2018-01-30 | 2021-03-18 | Genomatica, Inc. | Fermentation systems and methods with substantially uniform volumetric uptake rate of a reactive gaseous component |
| US11541105B2 (en) | 2018-06-01 | 2023-01-03 | The Research Foundation For The State University Of New York | Compositions and methods for disrupting biofilm formation and maintenance |
| CA3143683A1 (en) * | 2018-06-25 | 2020-01-02 | Lygos, Inc. | Recombinant host cells and methods for the production of aspartic acid and s-alanine |
| WO2020068900A1 (en) | 2018-09-26 | 2020-04-02 | Genomatica, Inc. | Aldehyde dehydrogenase variants and methods of using same |
| IT202000013243A1 (it) | 2020-06-04 | 2021-12-04 | Novamont Spa | Processo per la purificazione di una miscela di dioli |
| EP4419657A1 (en) * | 2021-10-20 | 2024-08-28 | Genomatica, Inc. | Aldehyde dehydrogenase variants and methods of use |
| IT202100030572A1 (it) | 2021-12-02 | 2023-06-02 | Novamont Spa | 1,3-butandiolo purificato da una miscela di dioli |
| WO2023140687A1 (ko) * | 2022-01-21 | 2023-07-27 | 대상 주식회사 | 1,4-부탄디올을 생산하는 미생물 및 이를 이용한 1,4-부탄디올의 생산 방법 |
| CN116083329A (zh) * | 2022-09-26 | 2023-05-09 | 北京绿色康成生物技术有限公司 | 发酵生产γ-丁内酯或1,4-丁二醇的方法 |
Citations (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2008115840A2 (en) * | 2007-03-16 | 2008-09-25 | Genomatica, Inc. | Compositions and methods for the biosynthesis of 1,4-butanediol and its precursors |
| WO2009111513A1 (en) * | 2008-03-03 | 2009-09-11 | Joule Biotechnologies, Inc. | Engineered co2 fixing microorganisms producing carbon-based products of interest |
Family Cites Families (207)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US4076948A (en) | 1968-10-10 | 1978-02-28 | El Paso Products Company | Process for treatment of adipic acid mother liquor |
| US3513209A (en) | 1968-08-19 | 1970-05-19 | Du Pont | Method of making 1,4-cyclohexadiene |
| GB1230276A (ja) | 1968-12-09 | 1971-04-28 | ||
| US3965182A (en) | 1969-10-02 | 1976-06-22 | Ethyl Corporation | Preparation of aniline from phenol and ammonia |
| JPS4831084B1 (ja) | 1970-09-04 | 1973-09-26 | ||
| GB1344557A (en) | 1972-06-23 | 1974-01-23 | Mitsubishi Petrochemical Co | Process for preparing 1,4-butanediol |
| JPS5543759B2 (ja) | 1972-06-28 | 1980-11-07 | ||
| DE2455617C3 (de) | 1974-11-23 | 1982-03-18 | Basf Ag, 6700 Ludwigshafen | Verfahren zur Herstellung von Butandiol und/oder Tetrahydrofuran über die Zwischenstufe des γ-Butyrolactons |
| DE2501499A1 (de) | 1975-01-16 | 1976-07-22 | Hoechst Ag | Verfahren zur herstellung von butandiol-(1.4) |
| US4190495A (en) | 1976-09-27 | 1980-02-26 | Research Corporation | Modified microorganisms and method of preparing and using same |
| GB1583517A (en) | 1977-05-04 | 1981-01-28 | Jackson J F | Solid bowl decanter centrifuges of the scroll discharge type |
| DE2842575A1 (de) * | 1977-10-04 | 1979-04-12 | Broadbent & Sons Ltd Thomas | Vollmantel-abklaerzentrifuge |
| JPS575693A (en) * | 1980-06-13 | 1982-01-12 | Ajinomoto Co Inc | Production of l-arginine through fermentation process |
| US4301077A (en) | 1980-12-22 | 1981-11-17 | Standard Oil Company | Process for the manufacture of 1-4-butanediol and tetrahydrofuran |
| JPS60114197A (ja) | 1983-11-25 | 1985-06-20 | Agency Of Ind Science & Technol | 微生物によるジカルボン酸の製造法 |
| US4871667A (en) | 1984-11-26 | 1989-10-03 | Agency Of Industrial Science & Technology | Process for preparing muconic acid |
| US4652685A (en) * | 1985-11-15 | 1987-03-24 | General Electric Company | Hydrogenation of lactones to glycols |
| US5168055A (en) | 1986-06-11 | 1992-12-01 | Rathin Datta | Fermentation and purification process for succinic acid |
| ES2036188T3 (es) | 1986-06-11 | 1993-05-16 | Michigan Biotechnology Institute | Un procedimiento para la produccion de acido succinico por fermentacion anaerobia. |
| US5143834A (en) | 1986-06-11 | 1992-09-01 | Glassner David A | Process for the production and purification of succinic acid |
| US5182199A (en) | 1987-05-27 | 1993-01-26 | Hartley Brian S | Thermophilic ethanol production in a two-stage closed system |
| US5245023A (en) * | 1987-06-29 | 1993-09-14 | Massachusetts Institute Of Technology | Method for producing novel polyester biopolymers |
| US5250430A (en) * | 1987-06-29 | 1993-10-05 | Massachusetts Institute Of Technology | Polyhydroxyalkanoate polymerase |
| US4876331A (en) * | 1987-08-18 | 1989-10-24 | Mitsubishi Kasei Corporation | Copolyester and process for producing the same |
| DE68923805T2 (de) | 1988-04-27 | 1995-12-07 | Daicel Chem | Verfahren zur herstellung von optisch aktivem 1,3-butanediol. |
| US4879420A (en) * | 1988-05-06 | 1989-11-07 | E.I. Du Pont De Nemours And Company | Preparation of mixtures of butanediols |
| EP0373230B1 (en) * | 1988-12-12 | 1993-02-17 | Unilever N.V. | Process for the microbiological preparation of 1,3-propane-diol from glycerol |
| CA2062753C (en) | 1989-04-27 | 2005-04-26 | N. Robert Ward, Jr. | Precipitate test for microorganisms |
| US5192673A (en) | 1990-04-30 | 1993-03-09 | Michigan Biotechnology Institute | Mutant strain of C. acetobutylicum and process for making butanol |
| US5079143A (en) | 1990-05-02 | 1992-01-07 | The Upjohn Company | Method of indentifying compounds useful as antiparasitic drugs |
| GB9011777D0 (en) * | 1990-05-25 | 1990-07-18 | Ici Plc | Hv/hb copolymer production |
| US5674978A (en) | 1990-09-21 | 1997-10-07 | The Regents Of The University Of California | Peptides derived from glutamic acid decarboxylase |
| US5475086A (en) * | 1990-09-21 | 1995-12-12 | The Regents Of The University Of California | Cloned glutamic acid decarboxylase peptides |
| US5998366A (en) | 1990-09-21 | 1999-12-07 | The Regents Of The University Of California | Method for ameliorating glutamic acid decarboxylase associated autoimmune disorders |
| US6682906B1 (en) * | 1990-09-21 | 2004-01-27 | The Regents Of The University Of California | Cloned glutamic acid decarboxylase |
| US5173429A (en) | 1990-11-09 | 1992-12-22 | The Board Of Trustees Of The University Of Arkansas | Clostridiumm ljungdahlii, an anaerobic ethanol and acetate producing microorganism |
| IL100572A (en) | 1991-01-03 | 1997-01-10 | Lepetit Spa | Amides of antibiotic ge 2270 factors their preparation and pharmaceutical compositions containing them |
| GB9108756D0 (en) * | 1991-04-24 | 1991-06-12 | Ici Plc | Production of polyalkanoate in plants |
| EP0522422A3 (en) * | 1991-07-01 | 1993-03-17 | Mitsubishi Kasei Corporation | Process for producing a biodegradable polymer |
| GB9115245D0 (en) * | 1991-07-16 | 1991-08-28 | Ici Plc | Production of polyalkanoate |
| US5610041A (en) * | 1991-07-19 | 1997-03-11 | Board Of Trustees Operating Michigan State University | Processes for producing polyhydroxybutyrate and related polyhydroxyalkanoates in the plastids of higher plants |
| ATE295891T1 (de) * | 1991-07-19 | 2005-06-15 | Univ Michigan State | Transgene pflanzen die polyhydroxyalkanoate produzieren |
| FR2680178B1 (fr) * | 1991-08-07 | 1994-10-14 | Ajinomoto Kk | Procede pour produire l'acide l-glutamique par fermentation. |
| JP2777757B2 (ja) * | 1991-09-17 | 1998-07-23 | 鐘淵化学工業株式会社 | 共重合体およびその製造方法 |
| DE4220759A1 (de) * | 1992-06-24 | 1994-01-05 | Inst Genbiologische Forschung | DNA-Sequenzen für Oligosaccharid-Transporter, Plasmide, Bakterien und Pflanzen enthaltend einen Transporter sowie Verfahren zur Herstellung und Transformation von Hefestämmen zur Identifikation des Transporteers |
| US5416020A (en) | 1992-09-29 | 1995-05-16 | Bio-Technical Resources | Lactobacillus delbrueckii ssp. bulgaricus strain and fermentation process for producing L-(+)-lactic acid |
| US6136577A (en) | 1992-10-30 | 2000-10-24 | Bioengineering Resources, Inc. | Biological production of ethanol from waste gases with Clostridium ljungdahlii |
| US5807722A (en) | 1992-10-30 | 1998-09-15 | Bioengineering Resources, Inc. | Biological production of acetic acid from waste gases with Clostridium ljungdahlii |
| WO1994012014A1 (en) * | 1992-11-20 | 1994-06-09 | Agracetus, Inc. | Transgenic cotton plants producing heterologous bioplastic |
| JP3263710B2 (ja) * | 1992-12-11 | 2002-03-11 | 高砂香料工業株式会社 | 生分解性光学活性ポリマー及びその製造方法 |
| FR2702492B1 (fr) | 1993-03-12 | 1995-05-24 | Rhone Poulenc Chimie | Procédé de production par fermentation d'acide itaconique. |
| JP3241505B2 (ja) * | 1993-08-11 | 2001-12-25 | 高砂香料工業株式会社 | 生分解性光学活性コポリマー及びその製造方法 |
| US5487987A (en) | 1993-09-16 | 1996-01-30 | Purdue Research Foundation | Synthesis of adipic acid from biomass-derived carbon sources |
| PL185681B1 (pl) * | 1993-10-28 | 2003-07-31 | Ajinomoto Kk | Sposób wytwarzania L-aminokwasu przy użyciu mikroorganizmu |
| GB9414506D0 (en) * | 1994-07-18 | 1994-09-07 | Zeneca Ltd | Improved production of polyhydroxyalkanoate |
| DE4433134A1 (de) * | 1994-09-16 | 1996-03-21 | Buck Chem Tech Werke | Verfahren zur Herstellung von Polyhydroxyfettsäuren sowie rekombinanter Bakterienstämme zur Durchführung des Verfahrens |
| US5563239A (en) * | 1994-11-09 | 1996-10-08 | Eastman Chemical Company | Composition and process for the production of poly(3-hydroxyalkanoates) |
| US5521075A (en) | 1994-12-19 | 1996-05-28 | Michigan Biotechnology Institute | Method for making succinic acid, anaerobiospirillum succiniciproducens variants for use in process and methods for obtaining variants |
| US5504004A (en) | 1994-12-20 | 1996-04-02 | Michigan Biotechnology Institute | Process for making succinic acid, microorganisms for use in the process and methods of obtaining the microorganisms |
| US5700934A (en) | 1995-03-01 | 1997-12-23 | Dsm N.V. | Process for the preparation of epsilon-caprolactam and epsilon-caprolactam precursors |
| US5478952A (en) | 1995-03-03 | 1995-12-26 | E. I. Du Pont De Nemours And Company | Ru,Re/carbon catalyst for hydrogenation in aqueous solution |
| US5863782A (en) | 1995-04-19 | 1999-01-26 | Women's And Children's Hospital | Synthetic mammalian sulphamidase and genetic sequences encoding same |
| US5686276A (en) | 1995-05-12 | 1997-11-11 | E. I. Du Pont De Nemours And Company | Bioconversion of a fermentable carbon source to 1,3-propanediol by a single microorganism |
| FR2736927B1 (fr) | 1995-07-18 | 1997-10-17 | Rhone Poulenc Fibres & Polymer | Enzymes a activite amidase, outils genetiques et microorganismes hotes permettant leur obtention et procede d'hydrolyse mettant en oeuvre lesdites enzymes |
| US5747311A (en) * | 1995-08-22 | 1998-05-05 | Microgen Corporation | Process for chemical modification of reactants by microbes |
| US5573931A (en) | 1995-08-28 | 1996-11-12 | Michigan Biotechnology Institute | Method for making succinic acid, bacterial variants for use in the process, and methods for obtaining variants |
| US5770435A (en) | 1995-11-02 | 1998-06-23 | University Of Chicago | Mutant E. coli strain with increased succinic acid production |
| US5869301A (en) | 1995-11-02 | 1999-02-09 | Lockhead Martin Energy Research Corporation | Method for the production of dicarboxylic acids |
| DE19543635A1 (de) * | 1995-11-23 | 1997-05-28 | Hp Chemie Pelzer Res & Dev | Verbundwerkstoffe aus Polyhydroxyfettsäuren und Fasermaterialien |
| US5849894A (en) | 1995-11-29 | 1998-12-15 | Monsanto Company | Rhodospirillum rubrum poly-β-hydroxyalkanoate synthase |
| AU2530797A (en) | 1996-02-27 | 1997-09-16 | Michigan State University | Cloning and expression of the gene encoding thermoanaerobacter ethanolicus 39E secondary-alcohol dehydrogenase and enzyme biochemical c haracterization |
| US6091002A (en) * | 1996-03-13 | 2000-07-18 | Monsanto Company | Polyhydroxyalkanoates of narrow molecular weight distribution prepared in transgenic plants |
| US5958745A (en) * | 1996-03-13 | 1999-09-28 | Monsanto Company | Methods of optimizing substrate pools and biosynthesis of poly-β-hydroxybutyrate-co-poly-β-hydroxyvalerate in bacteria and plants |
| US5959179A (en) * | 1996-03-13 | 1999-09-28 | Monsanto Company | Method for transforming soybeans |
| KR100387301B1 (ko) | 1996-07-01 | 2003-06-12 | 바이오 엔지니어링 리소스 인코포레이티드 | 폐가스로부터의 생성물의 생물학적 제조방법 |
| US5750848A (en) * | 1996-08-13 | 1998-05-12 | Monsanto Company | DNA sequence useful for the production of polyhydroxyalkanoates |
| KR100459818B1 (ko) | 1996-09-02 | 2004-12-03 | 이. 아이. 두퐁 드 느무르 앤드 컴퍼니 | ε-카프로락탐의 제조방법 |
| US6730503B1 (en) * | 1996-09-19 | 2004-05-04 | Roche Vitamins Inc. | Alcohol/aldehyde dehydrogenase |
| JPH10166664A (ja) * | 1996-12-12 | 1998-06-23 | Casio Electron Mfg Co Ltd | カラー印刷装置 |
| US6117658A (en) | 1997-02-13 | 2000-09-12 | James Madison University | Methods of making polyhydroxyalkanoates comprising 4-hydroxybutyrate monomer units |
| KR100516986B1 (ko) | 1997-02-19 | 2005-09-26 | 코닌클리즈케 디에스엠 엔.브이. | 6-아미노카프론산 유도체를 과열 수증기와 접촉시킴으로써 촉매 없이 카프로락탐을 제조하는 방법 |
| US6759219B2 (en) * | 1997-03-03 | 2004-07-06 | Metabolix, Inc. | Methods for the biosynthesis of polyesters |
| US6274790B1 (en) | 1997-04-14 | 2001-08-14 | The University Of British Columbia | Nucleic acids encoding a plant enzyme involved in very long chain fatty acid synthesis |
| US5994478A (en) * | 1997-04-21 | 1999-11-30 | Monsanto Company | Hydroxy-terminated polyhydroxyalkanoates |
| US6156852A (en) | 1997-04-21 | 2000-12-05 | Monsanto Company | Hydroxy-terminated polyhydroxyalkanoates |
| KR19990013007A (ko) * | 1997-07-31 | 1999-02-25 | 박원훈 | 형질전환된 대장균 ss373(kctc 8818p)과 이를 이용한숙신산의 생산방법 |
| DE69838768T2 (de) * | 1997-09-19 | 2008-10-30 | Metabolix, Inc., Cambridge | Biologische Systeme zur Herstellung von Polyhydroxyalkanoat-Polymeren die 4-Hy droxysäure enthalten |
| JPH11103863A (ja) | 1997-10-08 | 1999-04-20 | Nippon Shokubai Co Ltd | マレイン酸異性化酵素遺伝子 |
| US6228579B1 (en) * | 1997-11-14 | 2001-05-08 | San Diego State University Foundation | Method for identifying microbial proliferation genes |
| US6280986B1 (en) | 1997-12-01 | 2001-08-28 | The United States Of America As Represented By The Secretary Of Agriculture | Stabilization of pet operon plasmids and ethanol production in bacterial strains lacking lactate dehydrogenase and pyruvate formate lyase activities |
| WO1999053035A1 (en) | 1998-04-13 | 1999-10-21 | The University Of Georgia Research Foundation, Inc. | Pyruvate carboxylase overexpression for enhanced production of oxaloacetate-derived biochemicals in microbial cells |
| US20030087381A1 (en) | 1998-04-13 | 2003-05-08 | University Of Georgia Research Foundation, Inc. | Metabolically engineered organisms for enhanced production of oxaloacetate-derived biochemicals |
| US6159738A (en) | 1998-04-28 | 2000-12-12 | University Of Chicago | Method for construction of bacterial strains with increased succinic acid production |
| US6432686B1 (en) | 1998-05-12 | 2002-08-13 | E. I. Du Pont De Nemours And Company | Method for the production of 1,3-propanediol by recombinant organisms comprising genes for vitamin B12 transport |
| US6444784B1 (en) | 1998-05-29 | 2002-09-03 | Exxonmobil Research & Engineering Company | Wax crystal modifiers (LAW657) |
| WO2000004163A1 (en) * | 1998-07-15 | 2000-01-27 | E.I. Du Pont De Nemours And Company | Tetrahydrofolate metabolism enzymes |
| ES2302386T3 (es) * | 1998-08-04 | 2008-07-01 | Metabolix, Inc. | Produccion de polihidroxialcanoatos a partir de polioles. |
| ATE319835T1 (de) * | 1998-08-18 | 2006-03-15 | Metabolix Inc | Transgene polyhydroxyalkanoat produzierende mikroben |
| DE19856136C2 (de) | 1998-12-04 | 2002-10-24 | Pasteur Institut | Verfahren und Vorrichtung zur Selektion beschleunigter Proliferation lebender Zellen in Suspension |
| US6835820B2 (en) | 1999-01-07 | 2004-12-28 | The University Of Massachusetts | Polyhydroxyalkanoate biosynthesis associated proteins and coding region in bacillus megaterium |
| AU3482200A (en) | 1999-02-02 | 2000-08-25 | Bernhard Palsson | Methods for identifying drug targets based on genomic sequence data |
| US6686310B1 (en) | 1999-02-09 | 2004-02-03 | E. I. Du Pont De Nemours And Company | High surface area sol-gel route prepared hydrogenation catalysts |
| US6365376B1 (en) | 1999-02-19 | 2002-04-02 | E. I. Du Pont De Nemours And Company | Genes and enzymes for the production of adipic acid intermediates |
| WO2000052183A1 (en) | 1999-03-05 | 2000-09-08 | Monsanto Technology Llc | Multigene expression vectors for the biosynthesis of products via multienzyme biological pathways |
| KR100329019B1 (ko) * | 1999-04-13 | 2002-03-18 | 윤덕용 | 유기산의 고효율 생산방법 |
| WO2000071738A1 (en) | 1999-05-21 | 2000-11-30 | Cargill Dow Llc | Methods and materials for the synthesis of organic products |
| BR0013315B1 (pt) * | 1999-08-18 | 2013-06-25 | fragmento de Ácido nucleico isolado, polipeptÍdeo, gene quimÉrico, microorganismo, microorganismo recombinante , e, coli recombinante, linhagem klp23 de e. coli recombinante, linhagem rj8 de e. coli recombinante, vetor pdt29, vetor pkp32 e processos de bioproduÇço de 1,3-propanodiol | |
| US6852517B1 (en) | 1999-08-30 | 2005-02-08 | Wisconsin Alumni Research Foundation | Production of 3-hydroxypropionic acid in recombinant organisms |
| US6361983B1 (en) * | 1999-09-30 | 2002-03-26 | E. I. Du Pont De Nemours And Company | Process for the isolation of 1,3-propanediol from fermentation broth |
| US6897055B2 (en) * | 1999-11-25 | 2005-05-24 | Degussa Ag | Nucleotide sequences coding for the genes sucC and sucD |
| DE19956686A1 (de) * | 1999-11-25 | 2001-05-31 | Degussa | Neue für die Gene sucC und sucD codierende Nukleotidsequenzen |
| US6660857B2 (en) | 2000-02-03 | 2003-12-09 | Dsm N.V. | Process for the preparation of ε-caprolactam |
| US6878861B2 (en) | 2000-07-21 | 2005-04-12 | Washington State University Research Foundation | Acyl coenzyme A thioesterases |
| AU7599601A (en) * | 2000-07-21 | 2002-02-05 | Metabolix Inc | Production of polyhydroxyalkanoates from polyols |
| DK1303629T3 (da) | 2000-07-25 | 2006-10-30 | Emmaus Foundation Inc | Fremgangsmåde til forögelse af ethanolproduktion fra mikrobiel fermetering |
| WO2002016627A2 (en) | 2000-08-18 | 2002-02-28 | Tepha, Inc. | Sulfur containing polyhydroxyalkanoate compositions and method of production |
| US6916637B2 (en) * | 2000-09-30 | 2005-07-12 | Degussa Ag | Fermentation process for the preparation of L-amino acids using strains of the family Enterobacteriaceae |
| AU2002219818B2 (en) | 2000-11-20 | 2007-08-16 | Cargill, Incorporated | 3-hydroxypropionic acid and other organic compounds |
| AU2002237676A1 (en) | 2000-11-22 | 2002-06-03 | Cargill Dow Polymers, Llc | Methods and materials for the synthesis of organic products |
| CN1358841A (zh) | 2000-12-11 | 2002-07-17 | 云南省微生物研究所 | 云南链霉菌 |
| EP1354955A4 (en) | 2000-12-28 | 2005-01-05 | Toyota Motor Co Ltd | PROCESS FOR PREPARING PRENYL ALCOHOL |
| AU2002239855B2 (en) * | 2001-01-10 | 2006-11-23 | The Penn State Research Foundation | Method and system for modeling cellular metabolism |
| US7127379B2 (en) | 2001-01-31 | 2006-10-24 | The Regents Of The University Of California | Method for the evolutionary design of biochemical reaction networks |
| US20030059792A1 (en) | 2001-03-01 | 2003-03-27 | Palsson Bernhard O. | Models and methods for determining systemic properties of regulated reaction networks |
| US6743610B2 (en) | 2001-03-30 | 2004-06-01 | The University Of Chicago | Method to produce succinic acid from raw hydrolysates |
| US7052883B2 (en) * | 2001-04-03 | 2006-05-30 | Degussa Ag | Process for the production of L-amino acids using strains of the family Enterobacteriaceae that contain an attenuated fruR gene |
| WO2003008604A2 (en) * | 2001-07-18 | 2003-01-30 | Degussa Ag | Process for the preparation of l-amino acids using strains of the enterobacteriaceae family which contain an attenuated aceb gene |
| CA2356540A1 (en) | 2001-08-30 | 2003-02-28 | Emory University | Expressed dna sequences involved in mitochondrial functions |
| CA2466133A1 (en) | 2001-11-02 | 2003-05-15 | Rice University | Recycling system for manipulation of intracellular nadh availability |
| US20090158452A1 (en) * | 2001-12-04 | 2009-06-18 | Johnson Richard G | Transgenic plants with enhanced agronomic traits |
| US20090100536A1 (en) * | 2001-12-04 | 2009-04-16 | Monsanto Company | Transgenic plants with enhanced agronomic traits |
| CN1217001C (zh) * | 2002-01-04 | 2005-08-31 | 清华大学 | 一种生产d-(-)-3-羟基丁酸的方法 |
| MXPA04006894A (es) * | 2002-01-18 | 2004-10-15 | Cargill Inc | Alanina 2,3 - aminomutasa. |
| JPWO2003066863A1 (ja) | 2002-02-06 | 2005-06-02 | 昭和電工株式会社 | α−置換−α,β−不飽和カルボニル化合物の還元酵素遺伝子 |
| US7314974B2 (en) * | 2002-02-21 | 2008-01-01 | Monsanto Technology, Llc | Expression of microbial proteins in plants for production of plants with improved properties |
| US20030224363A1 (en) | 2002-03-19 | 2003-12-04 | Park Sung M. | Compositions and methods for modeling bacillus subtilis metabolism |
| JP2005521929A (ja) | 2002-03-29 | 2005-07-21 | ジェノマティカ・インコーポレイテッド | ヒト代謝モデルおよび方法 |
| US20050287655A1 (en) | 2002-05-10 | 2005-12-29 | Kyowa Hakko Kogyo Co., Ltd. | Process for producing mevalonic acid |
| US7856317B2 (en) | 2002-06-14 | 2010-12-21 | Genomatica, Inc. | Systems and methods for constructing genomic-based phenotypic models |
| AU2003256480B2 (en) | 2002-07-10 | 2008-03-06 | The Penn State Research Foundation | Method for determining gene knockout strategies |
| US7826975B2 (en) | 2002-07-10 | 2010-11-02 | The Penn State Research Foundation | Method for redesign of microbial production systems |
| WO2004007688A2 (en) | 2002-07-15 | 2004-01-22 | Kosan Biosciences, Inc. | Metabolic pathways for starter units in polyketide biosynthesis |
| BR0305767A (pt) * | 2002-08-09 | 2005-05-24 | Codexis Inc | Métodos e composições para a preparação de derivados de ácido 4-substituìdo 3-hidroxibutìrico |
| EP1581619B1 (en) * | 2002-09-12 | 2011-04-13 | Metabolix, Inc. | Polyhydroxyalkanoate production by coenzyme a-dependent aldehyde dehydrogenase pathways |
| EP1543114B1 (en) | 2002-09-27 | 2008-08-13 | DSM IP Assets B.V. | Aldehyde dehydrogenase gene |
| ATE493490T1 (de) | 2002-10-04 | 2011-01-15 | Du Pont | Verfahren zur biologischen herstellung von 1,3- propandiol mit hoher ausbeute |
| WO2004035009A2 (en) | 2002-10-15 | 2004-04-29 | The Regents Of The University Of California | Methods and systems to identify operational reaction pathways |
| AU2003287625A1 (en) | 2002-11-06 | 2004-06-03 | University Of Florida | Materials and methods for the efficient production of acetate and other products |
| WO2004074495A1 (ja) | 2003-02-24 | 2004-09-02 | Research Institute Of Innovative Technology For The Earth | 微生物による高効率水素製造方法 |
| US7056714B2 (en) * | 2003-03-11 | 2006-06-06 | University Of Iowa Research Foundation, Inc. | Carboxylic acid reductase polypeptide, nucleotide sequence encoding same and methods of use |
| RU2268300C2 (ru) | 2003-04-07 | 2006-01-20 | Закрытое акционерное общество "Научно-исследовательский институт Аджиномото-Генетика" | СПОСОБ ПОЛУЧЕНИЯ L-АМИНОКИСЛОТ С ИСПОЛЬЗОВАНИЕМ БАКТЕРИЙ, ОБЛАДАЮЩИХ ПОВЫШЕННОЙ ЭКСПРЕССИЕЙ ГЕНА pckA |
| AU2004266100A1 (en) * | 2003-08-11 | 2005-03-03 | Codexis, Inc. | Enzymatic processes for the production of 4-substituted 3-hydroxybutyric acid derivatives and vicinal cyano, hydroxy substituted carboxylic acid esters |
| US7927859B2 (en) | 2003-08-22 | 2011-04-19 | Rice University | High molar succinate yield bacteria by increasing the intracellular NADH availability |
| BRPI0414300A (pt) | 2003-09-17 | 2006-11-07 | Mitsubishi Chem Corp | método para produzir ácido orgánico não amino |
| US7244610B2 (en) | 2003-11-14 | 2007-07-17 | Rice University | Aerobic succinate production in bacteria |
| FR2864967B1 (fr) | 2004-01-12 | 2006-05-19 | Metabolic Explorer Sa | Microorganisme evolue pour la production de 1,2-propanediol |
| US7491520B2 (en) | 2004-01-19 | 2009-02-17 | Dsm Ip Assets B.V. | Biochemical synthesis of 6-amino caproic acid |
| US7608700B2 (en) | 2004-03-08 | 2009-10-27 | North Carolina State University | Lactobacillus acidophilus nucleic acid sequences encoding stress-related proteins and uses therefor |
| DE102004031177A1 (de) | 2004-06-29 | 2006-01-19 | Henkel Kgaa | Neue Geruchsstoffe bildende Genprodukte von Bacillus licheniformis und darauf aufbauende verbesserte biotechnologische Produktionsverfahren |
| EA010179B1 (ru) * | 2004-07-15 | 2008-06-30 | ДСМ АйПи АССЕТС Б.В. | Биохимический синтез 1,4-бутандиамина |
| US7262046B2 (en) | 2004-08-09 | 2007-08-28 | Rice University | Aerobic succinate production in bacteria |
| US7601858B2 (en) * | 2004-08-17 | 2009-10-13 | Gs Cleantech Corporation | Method of processing ethanol byproducts and related subsystems |
| EP1781797B1 (en) * | 2004-08-27 | 2016-10-19 | Rice University | Mutant e. coli strain with increased succinic acid production |
| GB0419424D0 (en) * | 2004-09-02 | 2004-10-06 | Viragen Scotland Ltd | Transgene optimisation |
| EP2434015B1 (en) | 2004-09-09 | 2013-11-20 | Research Institute Of Innovative Technology For The Earth | DNA fragment having promoter function |
| JP2008513023A (ja) | 2004-09-17 | 2008-05-01 | ライス ユニバーシティー | 高コハク酸生産細菌 |
| US7569380B2 (en) * | 2004-12-22 | 2009-08-04 | Rice University | Simultaneous anaerobic production of isoamyl acetate and succinic acid |
| JP2006204255A (ja) | 2005-01-31 | 2006-08-10 | Canon Inc | アセチル−CoAアシルトランスフェラーゼ遺伝子破壊ポリヒドロキシアルカノエート生産菌、またこれを利用したポリヒドロキシアルカノエート生産方法 |
| US20090068207A1 (en) | 2005-04-15 | 2009-03-12 | Vascular Biogenics Ltd. | Compositions Containing Beta 2-Glycoprotein I-Derived Peptides for the Prevention and/or Treatment of Vascular Disease |
| KR100679638B1 (ko) | 2005-08-19 | 2007-02-06 | 한국과학기술원 | 포메이트 디하이드로게나제 d 또는 e를 코딩하는 유전자로 형질전환된 미생물 및 이를 이용한 숙신산의 제조방법 |
| KR100676160B1 (ko) | 2005-08-19 | 2007-02-01 | 한국과학기술원 | 말릭효소를 코딩하는 유전자로 형질전환된 재조합 미생물 및 이를 이용한 숙신산의 제조방법 |
| CN101300356A (zh) | 2005-09-09 | 2008-11-05 | 基因组股份公司 | 用于琥珀酸盐之生长关联性生产的方法和生物 |
| US9297028B2 (en) | 2005-09-29 | 2016-03-29 | Butamax Advanced Biofuels Llc | Fermentive production of four carbon alcohols |
| NZ566406A (en) | 2005-10-26 | 2012-04-27 | Butamax Advanced Biofuels Llc | Fermentive production of four carbon alcohols |
| US20080274526A1 (en) | 2007-05-02 | 2008-11-06 | Bramucci Michael G | Method for the production of isobutanol |
| CA2650505A1 (en) | 2006-05-01 | 2008-02-14 | University Of Florida Research Foundation, Inc. | Ethanol production in non-recombinant hosts |
| US8962298B2 (en) | 2006-05-02 | 2015-02-24 | Butamax Advanced Biofuels Llc | Recombinant host cell comprising a diol dehydratase |
| US8017364B2 (en) | 2006-12-12 | 2011-09-13 | Butamax(Tm) Advanced Biofuels Llc | Solvent tolerant microorganisms |
| MX2009010154A (es) * | 2007-03-20 | 2009-10-12 | Univ Florida | Materiales y metodos para la produccion eficiente de succinato y malato. |
| US20090111154A1 (en) * | 2007-04-04 | 2009-04-30 | The Regents Of The University Of California | Butanol production by recombinant microorganisms |
| EP2147111A4 (en) | 2007-04-18 | 2010-06-23 | Gevo Inc | MANIPULATED MICROORGANISMS FOR THE MANUFACTURE OF ISOPROPANOL |
| CN101680007A (zh) * | 2007-04-18 | 2010-03-24 | 纳幕尔杜邦公司 | 使用高活性酮醇酸还原异构酶来发酵生产异丁醇 |
| US8426173B2 (en) | 2007-05-02 | 2013-04-23 | Butamax (Tm) Advanced Biofuels Llc | Method for the production of 1-butanol |
| US20080274522A1 (en) | 2007-05-02 | 2008-11-06 | Bramucci Michael G | Method for the production of 2-butanone |
| US8426174B2 (en) | 2007-05-02 | 2013-04-23 | Butamax(Tm) Advanced Biofuels Llc | Method for the production of 2-butanol |
| WO2008144060A2 (en) | 2007-05-17 | 2008-11-27 | Tetravitae Bioscience, Inc. | Methods and compositions for producing solvents |
| US20090023182A1 (en) * | 2007-07-18 | 2009-01-22 | Schilling Christophe H | Complementary metabolizing organisms and methods of making same |
| EP2017344A1 (en) | 2007-07-20 | 2009-01-21 | Nederlandse Organisatie voor toegepast- natuurwetenschappelijk onderzoek TNO | Production of itaconic acid |
| US7947483B2 (en) | 2007-08-10 | 2011-05-24 | Genomatica, Inc. | Methods and organisms for the growth-coupled production of 1,4-butanediol |
| WO2009049274A2 (en) | 2007-10-12 | 2009-04-16 | The Regents Of The University Of California | Microorganism engineered to produce isopropanol |
| EP2245137B1 (en) | 2008-01-22 | 2017-08-16 | Genomatica, Inc. | Methods and organisms for utilizing synthesis gas or other gaseous carbon sources and methanol |
| US20090246842A1 (en) * | 2008-02-15 | 2009-10-01 | Gevo, Inc. | Engineered microorganisms for producing propanol |
| CN103555643B (zh) | 2008-03-27 | 2016-08-10 | 基因组股份公司 | 用于产生己二酸和其他化合物的微生物 |
| BRPI0911759A2 (pt) | 2008-05-01 | 2019-09-24 | Genomatica Inc | microorganismo para a produção de ácido metacrílico |
| WO2009155382A1 (en) | 2008-06-17 | 2009-12-23 | Genomatica, Inc. | Microorganisms and methods for the biosynthesis of fumarate, malate, and acrylate |
| BRPI0915749A2 (pt) * | 2008-07-08 | 2018-07-10 | Opx Biotechnologies Inc | métodos, composições e sistemas para produção biossintética de 1,4-butanodiol |
| JP5912529B2 (ja) | 2008-09-10 | 2016-04-27 | ゲノマチカ, インク. | 1,4−ブタンジオールの生成のための微生物体 |
| US8354563B2 (en) | 2008-10-16 | 2013-01-15 | Maverick Biofuels, Inc. | Methods and apparatus for synthesis of alcohols from syngas |
| KR20110097951A (ko) | 2008-12-16 | 2011-08-31 | 게노마티카 인코포레이티드 | 합성가스와 다른 탄소원을 유용 제품으로 전환시키기 위한 미생물 및 방법 |
| EP3199511B1 (en) | 2009-06-04 | 2020-01-29 | Genomatica, Inc. | Process of separating components of a fermentation broth |
| CN102498215A (zh) * | 2009-06-04 | 2012-06-13 | 基因组股份公司 | 生产1,4-丁二醇的微生物和相关方法 |
| MX2012003025A (es) | 2009-09-09 | 2012-06-27 | Genomatica Inc | Microorganismos y metodos para la co-produccion de isopropanol con alcoholes, dioles y acidos primarios. |
| JP2013507145A (ja) | 2009-10-13 | 2013-03-04 | ゲノマチカ, インク. | 1,4−ブタンジオール、4−ヒドロキシブタナール、4−ヒドロキシブチリル−CoA、プトレシン及び関連化合物の生成のための微生物体並びに関連する方法 |
| US8530210B2 (en) | 2009-11-25 | 2013-09-10 | Genomatica, Inc. | Microorganisms and methods for the coproduction 1,4-butanediol and gamma-butyrolactone |
| CN109136161A (zh) * | 2009-12-10 | 2019-01-04 | 基因组股份公司 | 合成气或其他气态碳源和甲醇转化为1,3-丁二醇的方法和有机体 |
| US8048661B2 (en) | 2010-02-23 | 2011-11-01 | Genomatica, Inc. | Microbial organisms comprising exogenous nucleic acids encoding reductive TCA pathway enzymes |
| US8445244B2 (en) | 2010-02-23 | 2013-05-21 | Genomatica, Inc. | Methods for increasing product yields |
-
2010
- 2010-10-13 JP JP2012534339A patent/JP2013507145A/ja active Pending
- 2010-10-13 US US12/904,130 patent/US8377666B2/en active Active
- 2010-10-13 WO PCT/US2010/052570 patent/WO2011047101A1/en active Application Filing
- 2010-10-13 EP EP10824061.5A patent/EP2488652A4/en not_active Withdrawn
- 2010-10-13 MX MX2012004291A patent/MX2012004291A/es active IP Right Grant
- 2010-10-13 CN CN201080056425.4A patent/CN102762735B/zh active Active
- 2010-10-13 AU AU2010306785A patent/AU2010306785B2/en active Active
- 2010-10-13 CA CA2777459A patent/CA2777459A1/en not_active Abandoned
- 2010-10-13 KR KR1020127012122A patent/KR20120083908A/ko not_active Application Discontinuation
- 2010-10-13 CN CN201510982953.7A patent/CN105441374A/zh active Pending
-
2011
- 2011-05-17 US US13/109,732 patent/US8377667B2/en active Active
-
2012
- 2012-04-13 US US13/447,094 patent/US20130029381A1/en not_active Abandoned
- 2012-05-10 CO CO12077180A patent/CO6541567A2/es unknown
-
2013
- 2013-01-09 US US13/737,866 patent/US20130189751A1/en not_active Abandoned
-
2016
- 2016-06-03 US US15/173,427 patent/US20160376614A1/en not_active Abandoned
-
2018
- 2018-12-11 US US16/216,865 patent/US20190264240A1/en not_active Abandoned
Patent Citations (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2008115840A2 (en) * | 2007-03-16 | 2008-09-25 | Genomatica, Inc. | Compositions and methods for the biosynthesis of 1,4-butanediol and its precursors |
| WO2009111513A1 (en) * | 2008-03-03 | 2009-09-11 | Joule Biotechnologies, Inc. | Engineered co2 fixing microorganisms producing carbon-based products of interest |
Cited By (8)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| KR20160095053A (ko) * | 2013-12-05 | 2016-08-10 | 알이지 라이프 사이언시스, 엘엘씨 | 지방족 아민의 미생물 생성 |
| JP2016538870A (ja) * | 2013-12-05 | 2016-12-15 | アールイージー ライフ サイエンシズ リミテッド ライアビリティ カンパニー | 脂肪アミンの微生物生産方法 |
| KR102321813B1 (ko) * | 2013-12-05 | 2021-11-04 | 게노마티카 인코포레이티드 | 지방족 아민의 미생물 생성 |
| JP2021177785A (ja) * | 2013-12-05 | 2021-11-18 | ジェノマティカ, インコーポレイテッド | 脂肪アミンの微生物生産方法 |
| US11814660B2 (en) | 2013-12-05 | 2023-11-14 | Genomatica, Inc. | Recombinant microorganisms for the production of fatty amines |
| JP2021503306A (ja) * | 2017-11-17 | 2021-02-12 | エンザイミカルズ アーゲー | 塩沈殿下のトランスアミナーゼ反応によりカルボニル化合物からアミンを調製する方法 |
| JP7250807B2 (ja) | 2017-11-17 | 2023-04-03 | エンザイミカルズ アーゲー | 塩沈殿下のトランスアミナーゼ反応によりカルボニル化合物からアミンを調製する方法 |
| US12018303B2 (en) | 2017-11-17 | 2024-06-25 | Enzymicals Ag | Method for preparing amines from carbonyl compounds by transaminase reaction under salt precipitation |
Also Published As
| Publication number | Publication date |
|---|---|
| CN102762735A (zh) | 2012-10-31 |
| US20110129899A1 (en) | 2011-06-02 |
| CN105441374A (zh) | 2016-03-30 |
| MX2012004291A (es) | 2012-08-17 |
| US8377666B2 (en) | 2013-02-19 |
| CO6541567A2 (es) | 2012-10-16 |
| US20130029381A1 (en) | 2013-01-31 |
| WO2011047101A1 (en) | 2011-04-21 |
| US20110229946A1 (en) | 2011-09-22 |
| CA2777459A1 (en) | 2011-04-21 |
| EP2488652A1 (en) | 2012-08-22 |
| US20130189751A1 (en) | 2013-07-25 |
| US8377667B2 (en) | 2013-02-19 |
| CN102762735B (zh) | 2016-08-03 |
| EP2488652A4 (en) | 2014-04-09 |
| AU2010306785A1 (en) | 2012-04-26 |
| US20160376614A1 (en) | 2016-12-29 |
| KR20120083908A (ko) | 2012-07-26 |
| AU2010306785B2 (en) | 2016-01-14 |
| US20190264240A1 (en) | 2019-08-29 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| US20230065295A1 (en) | Microorganisms and methods for production of 4-hydroxybutyrate, 1,4-butanediol and related compounds | |
| US20190264240A1 (en) | Microorganisms for the production of 1,4-butanediol, 4-hydroxybutanal, 4-hydroxybutyryl-coa, putrescine and related compounds, and methods related thereto | |
| US10273508B2 (en) | Microorganisms for the production of 1,4-butanediol and related methods | |
| US20180142269A1 (en) | Microorganisms for the production of 1,4-butanediol | |
| AU2013203176A1 (en) | Microorganisms for the production of 1,4-butanediol,4-hydroxybutanal, 4-hydroxy-butyryl-coa, putrescine and related compounds, and methods related thereto | |
| AU2013203177B2 (en) | Microorganisms for the production of 1,4-butanediol and related methods | |
| AU2013203480A1 (en) | Microorganisms for the production of 1,4-butanediol |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20131011 |
|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20131011 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20150113 |
|
| A601 | Written request for extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A601 Effective date: 20150407 |
|
| A02 | Decision of refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A02 Effective date: 20151110 |