KR20180021137A - 키메라 항원 수용체 (car), 조성물 및 이의 사용 방법 - Google Patents
키메라 항원 수용체 (car), 조성물 및 이의 사용 방법 Download PDFInfo
- Publication number
- KR20180021137A KR20180021137A KR1020187002389A KR20187002389A KR20180021137A KR 20180021137 A KR20180021137 A KR 20180021137A KR 1020187002389 A KR1020187002389 A KR 1020187002389A KR 20187002389 A KR20187002389 A KR 20187002389A KR 20180021137 A KR20180021137 A KR 20180021137A
- Authority
- KR
- South Korea
- Prior art keywords
- cells
- cell
- gly
- leu
- ser
- Prior art date
Links
- 108010019670 Chimeric Antigen Receptors Proteins 0.000 title claims abstract description 392
- 238000000034 method Methods 0.000 title claims abstract description 76
- 239000000203 mixture Substances 0.000 title abstract description 14
- 108090000765 processed proteins & peptides Proteins 0.000 claims abstract description 156
- 102000004196 processed proteins & peptides Human genes 0.000 claims abstract description 130
- 229920001184 polypeptide Polymers 0.000 claims abstract description 116
- 239000003623 enhancer Substances 0.000 claims abstract description 30
- 210000004027 cell Anatomy 0.000 claims description 754
- 210000001744 T-lymphocyte Anatomy 0.000 claims description 188
- 239000000427 antigen Substances 0.000 claims description 188
- 108091007433 antigens Proteins 0.000 claims description 187
- 102000036639 antigens Human genes 0.000 claims description 187
- 210000000822 natural killer cell Anatomy 0.000 claims description 114
- 101000934338 Homo sapiens Myeloid cell surface antigen CD33 Proteins 0.000 claims description 74
- 102100025243 Myeloid cell surface antigen CD33 Human genes 0.000 claims description 74
- 101000738771 Homo sapiens Receptor-type tyrosine-protein phosphatase C Proteins 0.000 claims description 73
- 102100037422 Receptor-type tyrosine-protein phosphatase C Human genes 0.000 claims description 70
- 108090000623 proteins and genes Proteins 0.000 claims description 70
- 101000633784 Homo sapiens SLAM family member 7 Proteins 0.000 claims description 67
- 101000998120 Homo sapiens Interleukin-3 receptor subunit alpha Proteins 0.000 claims description 65
- 102100033493 Interleukin-3 receptor subunit alpha Human genes 0.000 claims description 65
- 239000013598 vector Substances 0.000 claims description 54
- 208000032839 leukemia Diseases 0.000 claims description 50
- 108091033319 polynucleotide Proteins 0.000 claims description 49
- 102000040430 polynucleotide Human genes 0.000 claims description 49
- 239000002157 polynucleotide Substances 0.000 claims description 49
- 102000003812 Interleukin-15 Human genes 0.000 claims description 48
- 108090000172 Interleukin-15 Proteins 0.000 claims description 48
- 102100024222 B-lymphocyte antigen CD19 Human genes 0.000 claims description 44
- 101000980825 Homo sapiens B-lymphocyte antigen CD19 Proteins 0.000 claims description 44
- 101000795167 Homo sapiens Tumor necrosis factor receptor superfamily member 13B Proteins 0.000 claims description 40
- 102100029675 Tumor necrosis factor receptor superfamily member 13B Human genes 0.000 claims description 40
- 206010035226 Plasma cell myeloma Diseases 0.000 claims description 36
- 102000004169 proteins and genes Human genes 0.000 claims description 35
- 102100034922 T-cell surface glycoprotein CD8 alpha chain Human genes 0.000 claims description 34
- 230000002147 killing effect Effects 0.000 claims description 34
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 claims description 33
- 238000011282 treatment Methods 0.000 claims description 32
- 102100031585 ADP-ribosyl cyclase/cyclic ADP-ribose hydrolase 1 Human genes 0.000 claims description 30
- 101000777636 Homo sapiens ADP-ribosyl cyclase/cyclic ADP-ribose hydrolase 1 Proteins 0.000 claims description 30
- 102100029690 Tumor necrosis factor receptor superfamily member 13C Human genes 0.000 claims description 28
- 101710178300 Tumor necrosis factor receptor superfamily member 13C Proteins 0.000 claims description 28
- 102100022005 B-lymphocyte antigen CD20 Human genes 0.000 claims description 26
- 101000897405 Homo sapiens B-lymphocyte antigen CD20 Proteins 0.000 claims description 26
- 101000716102 Homo sapiens T-cell surface glycoprotein CD4 Proteins 0.000 claims description 26
- 102100036011 T-cell surface glycoprotein CD4 Human genes 0.000 claims description 26
- 201000010099 disease Diseases 0.000 claims description 25
- 230000000295 complement effect Effects 0.000 claims description 24
- 101000874179 Homo sapiens Syndecan-1 Proteins 0.000 claims description 23
- 101000801255 Homo sapiens Tumor necrosis factor receptor superfamily member 17 Proteins 0.000 claims description 23
- 102100035721 Syndecan-1 Human genes 0.000 claims description 23
- 102100033726 Tumor necrosis factor receptor superfamily member 17 Human genes 0.000 claims description 23
- 239000012634 fragment Substances 0.000 claims description 23
- 210000004180 plasmocyte Anatomy 0.000 claims description 23
- 230000011664 signaling Effects 0.000 claims description 23
- 101000914514 Homo sapiens T-cell-specific surface glycoprotein CD28 Proteins 0.000 claims description 22
- 208000034578 Multiple myelomas Diseases 0.000 claims description 22
- 108010076504 Protein Sorting Signals Proteins 0.000 claims description 22
- 102100027213 T-cell-specific surface glycoprotein CD28 Human genes 0.000 claims description 22
- 238000003776 cleavage reaction Methods 0.000 claims description 22
- 102100038080 B-cell receptor CD22 Human genes 0.000 claims description 21
- 101000934341 Homo sapiens T-cell surface glycoprotein CD5 Proteins 0.000 claims description 21
- 101000851370 Homo sapiens Tumor necrosis factor receptor superfamily member 9 Proteins 0.000 claims description 21
- 102100025244 T-cell surface glycoprotein CD5 Human genes 0.000 claims description 21
- 102100036856 Tumor necrosis factor receptor superfamily member 9 Human genes 0.000 claims description 21
- 125000003275 alpha amino acid group Chemical group 0.000 claims description 20
- 230000007017 scission Effects 0.000 claims description 20
- 230000004936 stimulating effect Effects 0.000 claims description 20
- -1 LeY Proteins 0.000 claims description 19
- 102000013529 alpha-Fetoproteins Human genes 0.000 claims description 19
- 108010026331 alpha-Fetoproteins Proteins 0.000 claims description 19
- 101000884305 Homo sapiens B-cell receptor CD22 Proteins 0.000 claims description 17
- 102100020789 Interleukin-15 receptor subunit alpha Human genes 0.000 claims description 17
- 239000003446 ligand Substances 0.000 claims description 17
- 208000025324 B-cell acute lymphoblastic leukemia Diseases 0.000 claims description 16
- 102000005962 receptors Human genes 0.000 claims description 16
- 108020003175 receptors Proteins 0.000 claims description 16
- 102100025237 T-cell surface antigen CD2 Human genes 0.000 claims description 14
- 101710107699 Interleukin-15 receptor subunit alpha Proteins 0.000 claims description 13
- 208000032791 BCR-ABL1 positive chronic myelogenous leukemia Diseases 0.000 claims description 12
- 102100026122 High affinity immunoglobulin gamma Fc receptor I Human genes 0.000 claims description 12
- 101000913074 Homo sapiens High affinity immunoglobulin gamma Fc receptor I Proteins 0.000 claims description 12
- 101001078143 Homo sapiens Integrin alpha-IIb Proteins 0.000 claims description 12
- 101000934346 Homo sapiens T-cell surface antigen CD2 Proteins 0.000 claims description 12
- 102100025306 Integrin alpha-IIb Human genes 0.000 claims description 12
- 102100027208 T-cell antigen CD7 Human genes 0.000 claims description 12
- 230000002062 proliferating effect Effects 0.000 claims description 12
- 101000934372 Homo sapiens Macrosialin Proteins 0.000 claims description 11
- 101000914496 Homo sapiens T-cell antigen CD7 Proteins 0.000 claims description 11
- 102100025136 Macrosialin Human genes 0.000 claims description 11
- 208000010833 Chronic myeloid leukaemia Diseases 0.000 claims description 10
- 101001015004 Homo sapiens Integrin beta-3 Proteins 0.000 claims description 10
- 101001008874 Homo sapiens Mast/stem cell growth factor receptor Kit Proteins 0.000 claims description 10
- 102100032999 Integrin beta-3 Human genes 0.000 claims description 10
- 108010013709 Leukocyte Common Antigens Proteins 0.000 claims description 10
- 206010025323 Lymphomas Diseases 0.000 claims description 10
- 102100027754 Mast/stem cell growth factor receptor Kit Human genes 0.000 claims description 10
- 101100335081 Mus musculus Flt3 gene Proteins 0.000 claims description 10
- 102100022749 Aminopeptidase N Human genes 0.000 claims description 9
- 101000757160 Homo sapiens Aminopeptidase N Proteins 0.000 claims description 9
- 108010038501 Interleukin-6 Receptors Proteins 0.000 claims description 9
- 208000033761 Myelogenous Chronic BCR-ABL Positive Leukemia Diseases 0.000 claims description 9
- 102100022153 Tumor necrosis factor receptor superfamily member 4 Human genes 0.000 claims description 9
- 102100035248 Alpha-(1,3)-fucosyltransferase 4 Human genes 0.000 claims description 8
- 208000003950 B-cell lymphoma Diseases 0.000 claims description 8
- 102100025570 Cancer/testis antigen 1 Human genes 0.000 claims description 8
- 101001022185 Homo sapiens Alpha-(1,3)-fucosyltransferase 4 Proteins 0.000 claims description 8
- 101000856237 Homo sapiens Cancer/testis antigen 1 Proteins 0.000 claims description 8
- 101000946889 Homo sapiens Monocyte differentiation antigen CD14 Proteins 0.000 claims description 8
- 102100037792 Interleukin-6 receptor subunit alpha Human genes 0.000 claims description 8
- 102000017095 Leukocyte Common Antigens Human genes 0.000 claims description 8
- 102100035877 Monocyte differentiation antigen CD14 Human genes 0.000 claims description 8
- 101710089372 Programmed cell death protein 1 Proteins 0.000 claims description 8
- 101000851376 Homo sapiens Tumor necrosis factor receptor superfamily member 8 Proteins 0.000 claims description 7
- 102100036857 Tumor necrosis factor receptor superfamily member 8 Human genes 0.000 claims description 7
- 208000025750 heavy chain disease Diseases 0.000 claims description 7
- 101000916644 Homo sapiens Macrophage colony-stimulating factor 1 receptor Proteins 0.000 claims description 6
- 102100025390 Integrin beta-2 Human genes 0.000 claims description 6
- 102100028198 Macrophage colony-stimulating factor 1 receptor Human genes 0.000 claims description 6
- 101710165473 Tumor necrosis factor receptor superfamily member 4 Proteins 0.000 claims description 6
- 102000016605 B-Cell Activating Factor Human genes 0.000 claims description 5
- 108010028006 B-Cell Activating Factor Proteins 0.000 claims description 5
- 101000831616 Homo sapiens Protachykinin-1 Proteins 0.000 claims description 5
- 208000007452 Plasmacytoma Diseases 0.000 claims description 5
- 102100024304 Protachykinin-1 Human genes 0.000 claims description 5
- 206010042971 T-cell lymphoma Diseases 0.000 claims description 5
- 208000027585 T-cell non-Hodgkin lymphoma Diseases 0.000 claims description 5
- 230000001413 cellular effect Effects 0.000 claims description 5
- 208000010626 plasma cell neoplasm Diseases 0.000 claims description 5
- 230000019491 signal transduction Effects 0.000 claims description 5
- 102100023990 60S ribosomal protein L17 Human genes 0.000 claims description 4
- 108090000144 Human Proteins Proteins 0.000 claims description 4
- 102000003839 Human Proteins Human genes 0.000 claims description 4
- 206010020631 Hypergammaglobulinaemia benign monoclonal Diseases 0.000 claims description 4
- 208000019758 Hypergammaglobulinemia Diseases 0.000 claims description 4
- 102100030704 Interleukin-21 Human genes 0.000 claims description 4
- 239000003795 chemical substances by application Substances 0.000 claims description 4
- 230000007423 decrease Effects 0.000 claims description 4
- 108010074108 interleukin-21 Proteins 0.000 claims description 4
- 201000005328 monoclonal gammopathy of uncertain significance Diseases 0.000 claims description 4
- 230000037361 pathway Effects 0.000 claims description 4
- 208000031223 plasma cell leukemia Diseases 0.000 claims description 4
- 108010074708 B7-H1 Antigen Proteins 0.000 claims description 3
- 102100027207 CD27 antigen Human genes 0.000 claims description 3
- 102100038078 CD276 antigen Human genes 0.000 claims description 3
- 101710185679 CD276 antigen Proteins 0.000 claims description 3
- 101150013553 CD40 gene Proteins 0.000 claims description 3
- 101000914511 Homo sapiens CD27 antigen Proteins 0.000 claims description 3
- 101000935040 Homo sapiens Integrin beta-2 Proteins 0.000 claims description 3
- 102000037982 Immune checkpoint proteins Human genes 0.000 claims description 3
- 108091008036 Immune checkpoint proteins Proteins 0.000 claims description 3
- 108010064593 Intercellular Adhesion Molecule-1 Proteins 0.000 claims description 3
- 102100037877 Intercellular adhesion molecule 1 Human genes 0.000 claims description 3
- 108010064548 Lymphocyte Function-Associated Antigen-1 Proteins 0.000 claims description 3
- 101710160107 Outer membrane protein A Proteins 0.000 claims description 3
- 208000033014 Plasma cell tumor Diseases 0.000 claims description 3
- 102100024216 Programmed cell death 1 ligand 1 Human genes 0.000 claims description 3
- 102100040245 Tumor necrosis factor receptor superfamily member 5 Human genes 0.000 claims description 3
- 102100035793 CD83 antigen Human genes 0.000 claims description 2
- 101000946856 Homo sapiens CD83 antigen Proteins 0.000 claims description 2
- 101001109501 Homo sapiens NKG2-D type II integral membrane protein Proteins 0.000 claims description 2
- 101000830594 Homo sapiens Tumor necrosis factor ligand superfamily member 14 Proteins 0.000 claims description 2
- 102100022680 NKG2-D type II integral membrane protein Human genes 0.000 claims description 2
- 102100024586 Tumor necrosis factor ligand superfamily member 14 Human genes 0.000 claims description 2
- 239000003112 inhibitor Substances 0.000 claims description 2
- BGFTWECWAICPDG-UHFFFAOYSA-N 2-[bis(4-chlorophenyl)methyl]-4-n-[3-[bis(4-chlorophenyl)methyl]-4-(dimethylamino)phenyl]-1-n,1-n-dimethylbenzene-1,4-diamine Chemical compound C1=C(C(C=2C=CC(Cl)=CC=2)C=2C=CC(Cl)=CC=2)C(N(C)C)=CC=C1NC(C=1)=CC=C(N(C)C)C=1C(C=1C=CC(Cl)=CC=1)C1=CC=C(Cl)C=C1 BGFTWECWAICPDG-UHFFFAOYSA-N 0.000 claims 13
- 108010008014 B-Cell Maturation Antigen Proteins 0.000 claims 13
- 102000006942 B-Cell Maturation Antigen Human genes 0.000 claims 13
- 101001005269 Arabidopsis thaliana Ceramide synthase 1 LOH3 Proteins 0.000 claims 10
- 101001005312 Arabidopsis thaliana Ceramide synthase LOH1 Proteins 0.000 claims 10
- 102100028801 Calsyntenin-1 Human genes 0.000 claims 10
- 101000668858 Spinacia oleracea 30S ribosomal protein S1, chloroplastic Proteins 0.000 claims 10
- 101000898746 Streptomyces clavuligerus Clavaminate synthase 1 Proteins 0.000 claims 10
- 102100032530 Glypican-3 Human genes 0.000 claims 3
- 101001014668 Homo sapiens Glypican-3 Proteins 0.000 claims 3
- 210000000581 natural killer T-cell Anatomy 0.000 claims 3
- 102100034458 Hepatitis A virus cellular receptor 2 Human genes 0.000 claims 2
- 101710083479 Hepatitis A virus cellular receptor 2 homolog Proteins 0.000 claims 2
- 229940126547 T-cell immunoglobulin mucin-3 Drugs 0.000 claims 2
- 102100029360 Hematopoietic cell signal transducer Human genes 0.000 claims 1
- 101000990188 Homo sapiens Hematopoietic cell signal transducer Proteins 0.000 claims 1
- 101001109503 Homo sapiens NKG2-C type II integral membrane protein Proteins 0.000 claims 1
- 101000809875 Homo sapiens TYRO protein tyrosine kinase-binding protein Proteins 0.000 claims 1
- 102100022683 NKG2-C type II integral membrane protein Human genes 0.000 claims 1
- 102100038717 TYRO protein tyrosine kinase-binding protein Human genes 0.000 claims 1
- 208000033559 Waldenström macroglobulinemia Diseases 0.000 claims 1
- 210000000988 bone and bone Anatomy 0.000 claims 1
- 201000000564 macroglobulinemia Diseases 0.000 claims 1
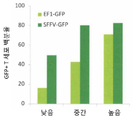
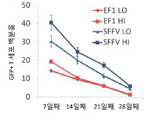
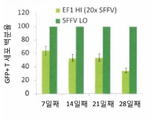
- 239000005090 green fluorescent protein Substances 0.000 description 115
- 108010043121 Green Fluorescent Proteins Proteins 0.000 description 95
- 102000004144 Green Fluorescent Proteins Human genes 0.000 description 95
- 230000014509 gene expression Effects 0.000 description 95
- 206010028980 Neoplasm Diseases 0.000 description 89
- 150000001875 compounds Chemical class 0.000 description 84
- 238000000684 flow cytometry Methods 0.000 description 60
- 102100029198 SLAM family member 7 Human genes 0.000 description 57
- 238000003501 co-culture Methods 0.000 description 48
- 201000011510 cancer Diseases 0.000 description 46
- 238000011534 incubation Methods 0.000 description 43
- 239000006228 supernatant Substances 0.000 description 42
- 206010057190 Respiratory tract infections Diseases 0.000 description 41
- 241000700605 Viruses Species 0.000 description 38
- 230000008685 targeting Effects 0.000 description 34
- 101000946843 Homo sapiens T-cell surface glycoprotein CD8 alpha chain Proteins 0.000 description 30
- 241000699670 Mus sp. Species 0.000 description 30
- 235000018102 proteins Nutrition 0.000 description 30
- 230000000694 effects Effects 0.000 description 28
- 102000017420 CD3 protein, epsilon/gamma/delta subunit Human genes 0.000 description 26
- 108050005493 CD3 protein, epsilon/gamma/delta subunit Proteins 0.000 description 26
- WSFSSNUMVMOOMR-UHFFFAOYSA-N Formaldehyde Chemical compound O=C WSFSSNUMVMOOMR-UHFFFAOYSA-N 0.000 description 26
- 239000012636 effector Substances 0.000 description 26
- 208000031261 Acute myeloid leukaemia Diseases 0.000 description 25
- 241000713880 Spleen focus-forming virus Species 0.000 description 25
- 238000002560 therapeutic procedure Methods 0.000 description 24
- 238000001890 transfection Methods 0.000 description 24
- 108091008874 T cell receptors Proteins 0.000 description 22
- 238000010361 transduction Methods 0.000 description 22
- 230000026683 transduction Effects 0.000 description 22
- 206010000830 Acute leukaemia Diseases 0.000 description 21
- 208000033776 Myeloid Acute Leukemia Diseases 0.000 description 20
- 102000016266 T-Cell Antigen Receptors Human genes 0.000 description 20
- 208000036676 acute undifferentiated leukemia Diseases 0.000 description 20
- 241000713666 Lentivirus Species 0.000 description 19
- 238000004458 analytical method Methods 0.000 description 19
- 102100031573 Hematopoietic progenitor cell antigen CD34 Human genes 0.000 description 18
- 101000777663 Homo sapiens Hematopoietic progenitor cell antigen CD34 Proteins 0.000 description 18
- 238000011198 co-culture assay Methods 0.000 description 18
- 230000003211 malignant effect Effects 0.000 description 16
- 241000124008 Mammalia Species 0.000 description 15
- 230000000875 corresponding effect Effects 0.000 description 15
- XKUKSGPZAADMRA-UHFFFAOYSA-N glycyl-glycyl-glycine Natural products NCC(=O)NCC(=O)NCC(O)=O XKUKSGPZAADMRA-UHFFFAOYSA-N 0.000 description 15
- 108091033409 CRISPR Proteins 0.000 description 14
- 210000003719 b-lymphocyte Anatomy 0.000 description 14
- 238000002474 experimental method Methods 0.000 description 14
- 230000001939 inductive effect Effects 0.000 description 14
- 201000000050 myeloid neoplasm Diseases 0.000 description 14
- 239000011886 peripheral blood Substances 0.000 description 14
- 210000005259 peripheral blood Anatomy 0.000 description 14
- 239000013612 plasmid Substances 0.000 description 14
- 230000004083 survival effect Effects 0.000 description 14
- 210000004881 tumor cell Anatomy 0.000 description 14
- 238000001262 western blot Methods 0.000 description 14
- 108010002350 Interleukin-2 Proteins 0.000 description 13
- 102000000588 Interleukin-2 Human genes 0.000 description 13
- 230000006037 cell lysis Effects 0.000 description 13
- 238000004090 dissolution Methods 0.000 description 13
- 239000013604 expression vector Substances 0.000 description 13
- 208000023275 Autoimmune disease Diseases 0.000 description 12
- 201000003793 Myelodysplastic syndrome Diseases 0.000 description 12
- 230000035755 proliferation Effects 0.000 description 12
- 241000699666 Mus <mouse, genus> Species 0.000 description 11
- 210000004700 fetal blood Anatomy 0.000 description 11
- 238000001727 in vivo Methods 0.000 description 11
- 208000002250 Hematologic Neoplasms Diseases 0.000 description 10
- 108010052781 Interleukin-3 Receptor alpha Subunit Proteins 0.000 description 10
- 102000018883 Interleukin-3 Receptor alpha Subunit Human genes 0.000 description 10
- 108020004684 Internal Ribosome Entry Sites Proteins 0.000 description 10
- 108700026244 Open Reading Frames Proteins 0.000 description 10
- 210000001185 bone marrow Anatomy 0.000 description 10
- 208000024908 graft versus host disease Diseases 0.000 description 10
- 238000000338 in vitro Methods 0.000 description 10
- 230000001965 increasing effect Effects 0.000 description 10
- 238000003780 insertion Methods 0.000 description 10
- 230000037431 insertion Effects 0.000 description 10
- 239000002773 nucleotide Substances 0.000 description 10
- 125000003729 nucleotide group Chemical group 0.000 description 10
- 230000003389 potentiating effect Effects 0.000 description 10
- 239000000047 product Substances 0.000 description 10
- 230000003612 virological effect Effects 0.000 description 10
- 108091007741 Chimeric antigen receptor T cells Proteins 0.000 description 9
- 208000009329 Graft vs Host Disease Diseases 0.000 description 9
- 230000003993 interaction Effects 0.000 description 9
- 102000006306 Antigen Receptors Human genes 0.000 description 8
- 108010083359 Antigen Receptors Proteins 0.000 description 8
- 108091027544 Subgenomic mRNA Proteins 0.000 description 8
- 230000000259 anti-tumor effect Effects 0.000 description 8
- 238000009169 immunotherapy Methods 0.000 description 8
- 230000002779 inactivation Effects 0.000 description 8
- 230000003834 intracellular effect Effects 0.000 description 8
- 230000004068 intracellular signaling Effects 0.000 description 8
- 239000002609 medium Substances 0.000 description 8
- 150000007523 nucleic acids Chemical class 0.000 description 8
- 239000013610 patient sample Substances 0.000 description 8
- RXWNCPJZOCPEPQ-NVWDDTSBSA-N puromycin Chemical compound C1=CC(OC)=CC=C1C[C@H](N)C(=O)N[C@H]1[C@@H](O)[C@H](N2C3=NC=NC(=C3N=C2)N(C)C)O[C@@H]1CO RXWNCPJZOCPEPQ-NVWDDTSBSA-N 0.000 description 8
- 238000012360 testing method Methods 0.000 description 8
- 210000001519 tissue Anatomy 0.000 description 8
- 239000013603 viral vector Substances 0.000 description 8
- HKZAAJSTFUZYTO-LURJTMIESA-N (2s)-2-[[2-[[2-[[2-[(2-aminoacetyl)amino]acetyl]amino]acetyl]amino]acetyl]amino]-3-hydroxypropanoic acid Chemical compound NCC(=O)NCC(=O)NCC(=O)NCC(=O)N[C@@H](CO)C(O)=O HKZAAJSTFUZYTO-LURJTMIESA-N 0.000 description 7
- 238000010354 CRISPR gene editing Methods 0.000 description 7
- 241000283707 Capra Species 0.000 description 7
- 108020004414 DNA Proteins 0.000 description 7
- SOEGEPHNZOISMT-BYPYZUCNSA-N Gly-Ser-Gly Chemical compound NCC(=O)N[C@@H](CO)C(=O)NCC(O)=O SOEGEPHNZOISMT-BYPYZUCNSA-N 0.000 description 7
- 108010029180 Sialic Acid Binding Ig-like Lectin 3 Proteins 0.000 description 7
- 102000001555 Sialic Acid Binding Ig-like Lectin 3 Human genes 0.000 description 7
- 108010003723 Single-Domain Antibodies Proteins 0.000 description 7
- KOSRFJWDECSPRO-UHFFFAOYSA-N alpha-L-glutamyl-L-glutamic acid Natural products OC(=O)CCC(N)C(=O)NC(CCC(O)=O)C(O)=O KOSRFJWDECSPRO-UHFFFAOYSA-N 0.000 description 7
- 210000000170 cell membrane Anatomy 0.000 description 7
- 238000002659 cell therapy Methods 0.000 description 7
- 230000009089 cytolysis Effects 0.000 description 7
- 230000006870 function Effects 0.000 description 7
- 102000037865 fusion proteins Human genes 0.000 description 7
- 108020001507 fusion proteins Proteins 0.000 description 7
- 108010055341 glutamyl-glutamic acid Proteins 0.000 description 7
- 238000004519 manufacturing process Methods 0.000 description 7
- 230000002688 persistence Effects 0.000 description 7
- 108010051242 phenylalanylserine Proteins 0.000 description 7
- 230000004044 response Effects 0.000 description 7
- 239000000523 sample Substances 0.000 description 7
- 102100024217 CAMPATH-1 antigen Human genes 0.000 description 6
- 108010065524 CD52 Antigen Proteins 0.000 description 6
- 102000004127 Cytokines Human genes 0.000 description 6
- 108090000695 Cytokines Proteins 0.000 description 6
- 102100041003 Glutamate carboxypeptidase 2 Human genes 0.000 description 6
- 101000892862 Homo sapiens Glutamate carboxypeptidase 2 Proteins 0.000 description 6
- 101001103039 Homo sapiens Inactive tyrosine-protein kinase transmembrane receptor ROR1 Proteins 0.000 description 6
- 101001103036 Homo sapiens Nuclear receptor ROR-alpha Proteins 0.000 description 6
- 241000701044 Human gammaherpesvirus 4 Species 0.000 description 6
- 241000701806 Human papillomavirus Species 0.000 description 6
- 108060003951 Immunoglobulin Proteins 0.000 description 6
- 102100039615 Inactive tyrosine-protein kinase transmembrane receptor ROR1 Human genes 0.000 description 6
- 230000004913 activation Effects 0.000 description 6
- 108010087924 alanylproline Proteins 0.000 description 6
- 230000000719 anti-leukaemic effect Effects 0.000 description 6
- 238000002512 chemotherapy Methods 0.000 description 6
- 230000001472 cytotoxic effect Effects 0.000 description 6
- 238000013461 design Methods 0.000 description 6
- 102000018358 immunoglobulin Human genes 0.000 description 6
- 210000000265 leukocyte Anatomy 0.000 description 6
- 210000004698 lymphocyte Anatomy 0.000 description 6
- 230000036210 malignancy Effects 0.000 description 6
- 239000003550 marker Substances 0.000 description 6
- 210000004379 membrane Anatomy 0.000 description 6
- 239000012528 membrane Substances 0.000 description 6
- 208000025113 myeloid leukemia Diseases 0.000 description 6
- 102000039446 nucleic acids Human genes 0.000 description 6
- 108020004707 nucleic acids Proteins 0.000 description 6
- 229920001481 poly(stearyl methacrylate) Polymers 0.000 description 6
- 230000001177 retroviral effect Effects 0.000 description 6
- 229930186217 Glycolipid Natural products 0.000 description 5
- 101000578784 Homo sapiens Melanoma antigen recognized by T-cells 1 Proteins 0.000 description 5
- 101001012157 Homo sapiens Receptor tyrosine-protein kinase erbB-2 Proteins 0.000 description 5
- WUHBLPVELFTPQK-KKUMJFAQSA-N Leu-Tyr-Asn Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(N)=O)C(O)=O WUHBLPVELFTPQK-KKUMJFAQSA-N 0.000 description 5
- 102100028389 Melanoma antigen recognized by T-cells 1 Human genes 0.000 description 5
- 102000000440 Melanoma-associated antigen Human genes 0.000 description 5
- 108050008953 Melanoma-associated antigen Proteins 0.000 description 5
- 102100030086 Receptor tyrosine-protein kinase erbB-2 Human genes 0.000 description 5
- 230000006044 T cell activation Effects 0.000 description 5
- 235000001014 amino acid Nutrition 0.000 description 5
- 238000013459 approach Methods 0.000 description 5
- 108010068380 arginylarginine Proteins 0.000 description 5
- 238000003556 assay Methods 0.000 description 5
- 108700010039 chimeric receptor Proteins 0.000 description 5
- 238000012258 culturing Methods 0.000 description 5
- 108010087914 epidermal growth factor receptor VIII Proteins 0.000 description 5
- MHMNJMPURVTYEJ-UHFFFAOYSA-N fluorescein-5-isothiocyanate Chemical compound O1C(=O)C2=CC(N=C=S)=CC=C2C21C1=CC=C(O)C=C1OC1=CC(O)=CC=C21 MHMNJMPURVTYEJ-UHFFFAOYSA-N 0.000 description 5
- 108010067216 glycyl-glycyl-glycine Proteins 0.000 description 5
- 108010089804 glycyl-threonine Proteins 0.000 description 5
- 108010057821 leucylproline Proteins 0.000 description 5
- 230000007246 mechanism Effects 0.000 description 5
- 238000012737 microarray-based gene expression Methods 0.000 description 5
- 238000012243 multiplex automated genomic engineering Methods 0.000 description 5
- 238000003752 polymerase chain reaction Methods 0.000 description 5
- 210000000130 stem cell Anatomy 0.000 description 5
- 230000001225 therapeutic effect Effects 0.000 description 5
- IASNWHAGGYTEKX-IUCAKERBSA-N Arg-Arg-Gly Chemical compound NC(N)=NCCC[C@H](N)C(=O)N[C@@H](CCCN=C(N)N)C(=O)NCC(O)=O IASNWHAGGYTEKX-IUCAKERBSA-N 0.000 description 4
- 206010006187 Breast cancer Diseases 0.000 description 4
- 208000026310 Breast neoplasm Diseases 0.000 description 4
- 208000035473 Communicable disease Diseases 0.000 description 4
- 241000701022 Cytomegalovirus Species 0.000 description 4
- 108010080611 Cytosine Deaminase Proteins 0.000 description 4
- 102000000311 Cytosine Deaminase Human genes 0.000 description 4
- IGXWBGJHJZYPQS-SSDOTTSWSA-N D-Luciferin Chemical compound OC(=O)[C@H]1CSC(C=2SC3=CC=C(O)C=C3N=2)=N1 IGXWBGJHJZYPQS-SSDOTTSWSA-N 0.000 description 4
- 229920000209 Hexadimethrine bromide Polymers 0.000 description 4
- 101001003140 Homo sapiens Interleukin-15 receptor subunit alpha Proteins 0.000 description 4
- IBMVEYRWAWIOTN-UHFFFAOYSA-N L-Leucyl-L-Arginyl-L-Proline Natural products CC(C)CC(N)C(=O)NC(CCCN=C(N)N)C(=O)N1CCCC1C(O)=O IBMVEYRWAWIOTN-UHFFFAOYSA-N 0.000 description 4
- LJBVRCDPWOJOEK-PPCPHDFISA-N Leu-Thr-Ile Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O LJBVRCDPWOJOEK-PPCPHDFISA-N 0.000 description 4
- SXOFUVGLPHCPRQ-KKUMJFAQSA-N Leu-Tyr-Cys Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CS)C(O)=O SXOFUVGLPHCPRQ-KKUMJFAQSA-N 0.000 description 4
- 108060001084 Luciferase Proteins 0.000 description 4
- 239000005089 Luciferase Substances 0.000 description 4
- YBAFDPFAUTYYRW-UHFFFAOYSA-N N-L-alpha-glutamyl-L-leucine Natural products CC(C)CC(C(O)=O)NC(=O)C(N)CCC(O)=O YBAFDPFAUTYYRW-UHFFFAOYSA-N 0.000 description 4
- 101710163270 Nuclease Proteins 0.000 description 4
- 108091028043 Nucleic acid sequence Proteins 0.000 description 4
- 206010033128 Ovarian cancer Diseases 0.000 description 4
- 208000006664 Precursor Cell Lymphoblastic Leukemia-Lymphoma Diseases 0.000 description 4
- 108010029157 Sialic Acid Binding Ig-like Lectin 2 Proteins 0.000 description 4
- APQIVBCUIUDSMB-OSUNSFLBSA-N Val-Ile-Thr Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H]([C@@H](C)O)C(=O)O)NC(=O)[C@H](C(C)C)N APQIVBCUIUDSMB-OSUNSFLBSA-N 0.000 description 4
- 108010081404 acein-2 Proteins 0.000 description 4
- 108010076324 alanyl-glycyl-glycine Proteins 0.000 description 4
- 108010086434 alanyl-seryl-glycine Proteins 0.000 description 4
- 150000001413 amino acids Chemical class 0.000 description 4
- 108010047857 aspartylglycine Proteins 0.000 description 4
- 230000022534 cell killing Effects 0.000 description 4
- 238000012512 characterization method Methods 0.000 description 4
- 231100000433 cytotoxic Toxicity 0.000 description 4
- 230000002950 deficient Effects 0.000 description 4
- 230000008030 elimination Effects 0.000 description 4
- 238000003379 elimination reaction Methods 0.000 description 4
- 238000001943 fluorescence-activated cell sorting Methods 0.000 description 4
- 238000001415 gene therapy Methods 0.000 description 4
- 108010050848 glycylleucine Proteins 0.000 description 4
- 230000012010 growth Effects 0.000 description 4
- 210000003958 hematopoietic stem cell Anatomy 0.000 description 4
- 238000003384 imaging method Methods 0.000 description 4
- 210000002865 immune cell Anatomy 0.000 description 4
- 230000028993 immune response Effects 0.000 description 4
- 108010003700 lysyl aspartic acid Proteins 0.000 description 4
- 230000009826 neoplastic cell growth Effects 0.000 description 4
- 238000011275 oncology therapy Methods 0.000 description 4
- 229950010131 puromycin Drugs 0.000 description 4
- 108010026333 seryl-proline Proteins 0.000 description 4
- 230000001988 toxicity Effects 0.000 description 4
- 231100000419 toxicity Toxicity 0.000 description 4
- 230000005740 tumor formation Effects 0.000 description 4
- HVCOBJNICQPDBP-UHFFFAOYSA-N 3-[3-[3,5-dihydroxy-6-methyl-4-(3,4,5-trihydroxy-6-methyloxan-2-yl)oxyoxan-2-yl]oxydecanoyloxy]decanoic acid;hydrate Chemical compound O.OC1C(OC(CC(=O)OC(CCCCCCC)CC(O)=O)CCCCCCC)OC(C)C(O)C1OC1C(O)C(O)C(O)C(C)O1 HVCOBJNICQPDBP-UHFFFAOYSA-N 0.000 description 3
- 208000024893 Acute lymphoblastic leukemia Diseases 0.000 description 3
- OQCWXQJLCDPRHV-UWVGGRQHSA-N Arg-Gly-Leu Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)NCC(=O)N[C@@H](CC(C)C)C(O)=O OQCWXQJLCDPRHV-UWVGGRQHSA-N 0.000 description 3
- LXMKTIZAGIBQRX-HRCADAONSA-N Arg-Phe-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CC2=CC=CC=C2)NC(=O)[C@H](CCCN=C(N)N)N)C(=O)O LXMKTIZAGIBQRX-HRCADAONSA-N 0.000 description 3
- WOZDCBHUGJVJPL-AVGNSLFASA-N Arg-Val-Lys Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CCCN=C(N)N)N WOZDCBHUGJVJPL-AVGNSLFASA-N 0.000 description 3
- PBVLJOIPOGUQQP-CIUDSAMLSA-N Asp-Ala-Leu Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](C)C(=O)N[C@@H](CC(C)C)C(O)=O PBVLJOIPOGUQQP-CIUDSAMLSA-N 0.000 description 3
- GYWQGGUCMDCUJE-DLOVCJGASA-N Asp-Phe-Ala Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](C)C(O)=O GYWQGGUCMDCUJE-DLOVCJGASA-N 0.000 description 3
- XMKXONRMGJXCJV-LAEOZQHASA-N Asp-Val-Glu Chemical compound OC(=O)C[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(O)=O)C(O)=O XMKXONRMGJXCJV-LAEOZQHASA-N 0.000 description 3
- 238000011357 CAR T-cell therapy Methods 0.000 description 3
- 241000282693 Cercopithecidae Species 0.000 description 3
- 206010009944 Colon cancer Diseases 0.000 description 3
- BCWIFCLVCRAIQK-ZLUOBGJFSA-N Cys-Ser-Cys Chemical compound C([C@@H](C(=O)N[C@@H](CS)C(=O)O)NC(=O)[C@H](CS)N)O BCWIFCLVCRAIQK-ZLUOBGJFSA-N 0.000 description 3
- 239000006144 Dulbecco’s modified Eagle's medium Substances 0.000 description 3
- XZLLTYBONVKGLO-SDDRHHMPSA-N Gln-Lys-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CCCCN)NC(=O)[C@H](CCC(=O)N)N)C(=O)O XZLLTYBONVKGLO-SDDRHHMPSA-N 0.000 description 3
- LXAUHIRMWXQRKI-XHNCKOQMSA-N Glu-Asn-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CC(=O)N)NC(=O)[C@H](CCC(=O)O)N)C(=O)O LXAUHIRMWXQRKI-XHNCKOQMSA-N 0.000 description 3
- DSPQRJXOIXHOHK-WDSKDSINSA-N Glu-Asp-Gly Chemical compound OC(=O)CC[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)NCC(O)=O DSPQRJXOIXHOHK-WDSKDSINSA-N 0.000 description 3
- UPOJUWHGMDJUQZ-IUCAKERBSA-N Gly-Arg-Arg Chemical compound NC(=N)NCCC[C@H](NC(=O)CN)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O UPOJUWHGMDJUQZ-IUCAKERBSA-N 0.000 description 3
- OCPPBNKYGYSLOE-IUCAKERBSA-N Gly-Pro-Met Chemical compound CSCC[C@@H](C(O)=O)NC(=O)[C@@H]1CCCN1C(=O)CN OCPPBNKYGYSLOE-IUCAKERBSA-N 0.000 description 3
- ZKJZBRHRWKLVSJ-ZDLURKLDSA-N Gly-Thr-Cys Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CS)C(=O)O)NC(=O)CN)O ZKJZBRHRWKLVSJ-ZDLURKLDSA-N 0.000 description 3
- 208000017604 Hodgkin disease Diseases 0.000 description 3
- 208000021519 Hodgkin lymphoma Diseases 0.000 description 3
- 208000010747 Hodgkins lymphoma Diseases 0.000 description 3
- 101000581981 Homo sapiens Neural cell adhesion molecule 1 Proteins 0.000 description 3
- 241000725303 Human immunodeficiency virus Species 0.000 description 3
- LWWILHPVAKKLQS-QXEWZRGKSA-N Ile-Gly-Met Chemical compound CC[C@H](C)[C@@H](C(=O)NCC(=O)N[C@@H](CCSC)C(=O)O)N LWWILHPVAKKLQS-QXEWZRGKSA-N 0.000 description 3
- 108010065920 Insulin Lispro Proteins 0.000 description 3
- 108010017535 Interleukin-15 Receptors Proteins 0.000 description 3
- 102000004556 Interleukin-15 Receptors Human genes 0.000 description 3
- 102000015696 Interleukins Human genes 0.000 description 3
- 108010063738 Interleukins Proteins 0.000 description 3
- IBMVEYRWAWIOTN-RWMBFGLXSA-N Leu-Arg-Pro Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CCCN=C(N)N)C(=O)N1CCC[C@@H]1C(O)=O IBMVEYRWAWIOTN-RWMBFGLXSA-N 0.000 description 3
- NRFGTHFONZYFNY-MGHWNKPDSA-N Leu-Ile-Tyr Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 NRFGTHFONZYFNY-MGHWNKPDSA-N 0.000 description 3
- RXGLHDWAZQECBI-SRVKXCTJSA-N Leu-Leu-Ser Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(O)=O RXGLHDWAZQECBI-SRVKXCTJSA-N 0.000 description 3
- 102100029193 Low affinity immunoglobulin gamma Fc region receptor III-A Human genes 0.000 description 3
- 108700018351 Major Histocompatibility Complex Proteins 0.000 description 3
- 102100027347 Neural cell adhesion molecule 1 Human genes 0.000 description 3
- 101100342977 Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) leu-1 gene Proteins 0.000 description 3
- 206010061535 Ovarian neoplasm Diseases 0.000 description 3
- WEDZFLRYSIDIRX-IHRRRGAJSA-N Phe-Ser-Arg Chemical compound NC(=N)NCCC[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CC1=CC=CC=C1 WEDZFLRYSIDIRX-IHRRRGAJSA-N 0.000 description 3
- LCRSGSIRKLXZMZ-BPNCWPANSA-N Pro-Ala-Tyr Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O LCRSGSIRKLXZMZ-BPNCWPANSA-N 0.000 description 3
- FMLRRBDLBJLJIK-DCAQKATOSA-N Pro-Leu-Ala Chemical compound OC(=O)[C@H](C)NC(=O)[C@H](CC(C)C)NC(=O)[C@@H]1CCCN1 FMLRRBDLBJLJIK-DCAQKATOSA-N 0.000 description 3
- FKYKZHOKDOPHSA-DCAQKATOSA-N Pro-Leu-Ser Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(O)=O FKYKZHOKDOPHSA-DCAQKATOSA-N 0.000 description 3
- YDTUEBLEAVANFH-RCWTZXSCSA-N Pro-Val-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)[C@H](C(C)C)NC(=O)[C@@H]1CCCN1 YDTUEBLEAVANFH-RCWTZXSCSA-N 0.000 description 3
- 208000006265 Renal cell carcinoma Diseases 0.000 description 3
- FMDHKPRACUXATF-ACZMJKKPSA-N Ser-Gln-Ser Chemical compound OC[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CO)C(O)=O FMDHKPRACUXATF-ACZMJKKPSA-N 0.000 description 3
- YMTLKLXDFCSCNX-BYPYZUCNSA-N Ser-Gly-Gly Chemical compound OC[C@H](N)C(=O)NCC(=O)NCC(O)=O YMTLKLXDFCSCNX-BYPYZUCNSA-N 0.000 description 3
- UIGMAMGZOJVTDN-WHFBIAKZSA-N Ser-Gly-Ser Chemical compound OC[C@H](N)C(=O)NCC(=O)N[C@@H](CO)C(O)=O UIGMAMGZOJVTDN-WHFBIAKZSA-N 0.000 description 3
- OQPNSDWGAMFJNU-QWRGUYRKSA-N Ser-Gly-Tyr Chemical compound OC[C@H](N)C(=O)NCC(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 OQPNSDWGAMFJNU-QWRGUYRKSA-N 0.000 description 3
- VUVCRYXYUUPGSB-GLLZPBPUSA-N Thr-Gln-Glu Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)N[C@@H](CCC(=O)O)C(=O)O)N)O VUVCRYXYUUPGSB-GLLZPBPUSA-N 0.000 description 3
- WTMPKZWHRCMMMT-KZVJFYERSA-N Thr-Pro-Ala Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N1CCC[C@H]1C(=O)N[C@@H](C)C(O)=O WTMPKZWHRCMMMT-KZVJFYERSA-N 0.000 description 3
- SMLCYZYQFRTLCO-UWJYBYFXSA-N Tyr-Cys-Ala Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CS)C(=O)N[C@@H](C)C(O)=O SMLCYZYQFRTLCO-UWJYBYFXSA-N 0.000 description 3
- ZIGZPYJXIWLQFC-QTKMDUPCSA-N Val-His-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CC1=CN=CN1)NC(=O)[C@H](C(C)C)N)O ZIGZPYJXIWLQFC-QTKMDUPCSA-N 0.000 description 3
- 230000002159 abnormal effect Effects 0.000 description 3
- 230000003044 adaptive effect Effects 0.000 description 3
- 108010008685 alanyl-glutamyl-aspartic acid Proteins 0.000 description 3
- 108010039538 alanyl-glycyl-aspartyl-valine Proteins 0.000 description 3
- 108010047495 alanylglycine Proteins 0.000 description 3
- 230000000735 allogeneic effect Effects 0.000 description 3
- 206010002022 amyloidosis Diseases 0.000 description 3
- 108010069926 arginyl-glycyl-serine Proteins 0.000 description 3
- 108010029539 arginyl-prolyl-proline Proteins 0.000 description 3
- 108010060035 arginylproline Proteins 0.000 description 3
- 108010077245 asparaginyl-proline Proteins 0.000 description 3
- 210000004369 blood Anatomy 0.000 description 3
- 239000008280 blood Substances 0.000 description 3
- 208000029742 colonic neoplasm Diseases 0.000 description 3
- 239000002299 complementary DNA Substances 0.000 description 3
- 230000003750 conditioning effect Effects 0.000 description 3
- 108010016616 cysteinylglycine Proteins 0.000 description 3
- 210000001151 cytotoxic T lymphocyte Anatomy 0.000 description 3
- 231100000673 dose–response relationship Toxicity 0.000 description 3
- 230000003828 downregulation Effects 0.000 description 3
- 210000001723 extracellular space Anatomy 0.000 description 3
- 239000012737 fresh medium Substances 0.000 description 3
- 230000002538 fungal effect Effects 0.000 description 3
- 230000004927 fusion Effects 0.000 description 3
- 108010078144 glutaminyl-glycine Proteins 0.000 description 3
- VPZXBVLAVMBEQI-UHFFFAOYSA-N glycyl-DL-alpha-alanine Natural products OC(=O)C(C)NC(=O)CN VPZXBVLAVMBEQI-UHFFFAOYSA-N 0.000 description 3
- 108010001064 glycyl-glycyl-glycyl-glycine Proteins 0.000 description 3
- 108010015792 glycyllysine Proteins 0.000 description 3
- 210000002443 helper t lymphocyte Anatomy 0.000 description 3
- 238000011134 hematopoietic stem cell transplantation Methods 0.000 description 3
- 238000003119 immunoblot Methods 0.000 description 3
- 238000002347 injection Methods 0.000 description 3
- 239000007924 injection Substances 0.000 description 3
- 108010073472 leucyl-prolyl-proline Proteins 0.000 description 3
- 230000000670 limiting effect Effects 0.000 description 3
- 108010057952 lysyl-phenylalanyl-lysine Proteins 0.000 description 3
- 230000001404 mediated effect Effects 0.000 description 3
- 201000001441 melanoma Diseases 0.000 description 3
- 210000003071 memory t lymphocyte Anatomy 0.000 description 3
- 239000002245 particle Substances 0.000 description 3
- MXHCPCSDRGLRER-UHFFFAOYSA-N pentaglycine Chemical compound NCC(=O)NCC(=O)NCC(=O)NCC(=O)NCC(O)=O MXHCPCSDRGLRER-UHFFFAOYSA-N 0.000 description 3
- 210000003819 peripheral blood mononuclear cell Anatomy 0.000 description 3
- 108010070409 phenylalanyl-glycyl-glycine Proteins 0.000 description 3
- 210000004986 primary T-cell Anatomy 0.000 description 3
- 230000008569 process Effects 0.000 description 3
- 108010070643 prolylglutamic acid Proteins 0.000 description 3
- 230000005855 radiation Effects 0.000 description 3
- 230000009467 reduction Effects 0.000 description 3
- 230000002829 reductive effect Effects 0.000 description 3
- 108010056030 retronectin Proteins 0.000 description 3
- 238000011476 stem cell transplantation Methods 0.000 description 3
- 230000020382 suppression by virus of host antigen processing and presentation of peptide antigen via MHC class I Effects 0.000 description 3
- 230000002459 sustained effect Effects 0.000 description 3
- 108010038745 tryptophylglycine Proteins 0.000 description 3
- 108010017949 tyrosyl-glycyl-glycine Proteins 0.000 description 3
- 241001430294 unidentified retrovirus Species 0.000 description 3
- 108010073969 valyllysine Proteins 0.000 description 3
- 108010000998 wheylin-2 peptide Proteins 0.000 description 3
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N (+)-Biotin Chemical compound N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 description 2
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 2
- PIDRBUDUWHBYSR-UHFFFAOYSA-N 1-[2-[[2-[(2-amino-4-methylpentanoyl)amino]-4-methylpentanoyl]amino]-4-methylpentanoyl]pyrrolidine-2-carboxylic acid Chemical compound CC(C)CC(N)C(=O)NC(CC(C)C)C(=O)NC(CC(C)C)C(=O)N1CCCC1C(O)=O PIDRBUDUWHBYSR-UHFFFAOYSA-N 0.000 description 2
- 208000014697 Acute lymphocytic leukaemia Diseases 0.000 description 2
- KXEVYGKATAMXJJ-ACZMJKKPSA-N Ala-Glu-Asp Chemical compound C[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(O)=O)C(O)=O KXEVYGKATAMXJJ-ACZMJKKPSA-N 0.000 description 2
- QQACQIHVWCVBBR-GVARAGBVSA-N Ala-Ile-Tyr Chemical compound [H]N[C@@H](C)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O QQACQIHVWCVBBR-GVARAGBVSA-N 0.000 description 2
- OYJCVIGKMXUVKB-GARJFASQSA-N Ala-Leu-Pro Chemical compound C[C@@H](C(=O)N[C@@H](CC(C)C)C(=O)N1CCC[C@@H]1C(=O)O)N OYJCVIGKMXUVKB-GARJFASQSA-N 0.000 description 2
- MAZZQZWCCYJQGZ-GUBZILKMSA-N Ala-Pro-Arg Chemical compound [H]N[C@@H](C)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CCCNC(N)=N)C(O)=O MAZZQZWCCYJQGZ-GUBZILKMSA-N 0.000 description 2
- FFZJHQODAYHGPO-KZVJFYERSA-N Ala-Pro-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)[C@@H]1CCCN1C(=O)[C@H](C)N FFZJHQODAYHGPO-KZVJFYERSA-N 0.000 description 2
- YYAVDNKUWLAFCV-ACZMJKKPSA-N Ala-Ser-Gln Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CO)C(=O)N[C@@H](CCC(N)=O)C(O)=O YYAVDNKUWLAFCV-ACZMJKKPSA-N 0.000 description 2
- SAHQGRZIQVEJPF-JXUBOQSCSA-N Ala-Thr-Lys Chemical compound C[C@H](N)C(=O)N[C@@H]([C@H](O)C)C(=O)N[C@H](C(O)=O)CCCCN SAHQGRZIQVEJPF-JXUBOQSCSA-N 0.000 description 2
- QRIYOHQJRDHFKF-UWJYBYFXSA-N Ala-Tyr-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)[C@@H](N)C)CC1=CC=C(O)C=C1 QRIYOHQJRDHFKF-UWJYBYFXSA-N 0.000 description 2
- GIVATXIGCXFQQA-FXQIFTODSA-N Arg-Ala-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@H](C)NC(=O)[C@@H](N)CCCN=C(N)N GIVATXIGCXFQQA-FXQIFTODSA-N 0.000 description 2
- MFAMTAVAFBPXDC-LPEHRKFASA-N Arg-Asp-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CC(=O)O)NC(=O)[C@H](CCCN=C(N)N)N)C(=O)O MFAMTAVAFBPXDC-LPEHRKFASA-N 0.000 description 2
- HQIZDMIGUJOSNI-IUCAKERBSA-N Arg-Gly-Arg Chemical compound N[C@@H](CCCNC(N)=N)C(=O)NCC(=O)N[C@@H](CCCNC(N)=N)C(O)=O HQIZDMIGUJOSNI-IUCAKERBSA-N 0.000 description 2
- WVNFNPGXYADPPO-BQBZGAKWSA-N Arg-Gly-Ser Chemical compound NC(N)=NCCC[C@H](N)C(=O)NCC(=O)N[C@@H](CO)C(O)=O WVNFNPGXYADPPO-BQBZGAKWSA-N 0.000 description 2
- SSZGOKWBHLOCHK-DCAQKATOSA-N Arg-Lys-Asn Chemical compound NC(=O)C[C@@H](C(O)=O)NC(=O)[C@H](CCCCN)NC(=O)[C@@H](N)CCCN=C(N)N SSZGOKWBHLOCHK-DCAQKATOSA-N 0.000 description 2
- DIIGDGJKTMLQQW-IHRRRGAJSA-N Arg-Lys-His Chemical compound C1=C(NC=N1)C[C@@H](C(=O)O)NC(=O)[C@H](CCCCN)NC(=O)[C@H](CCCN=C(N)N)N DIIGDGJKTMLQQW-IHRRRGAJSA-N 0.000 description 2
- OVQJAKFLFTZDNC-GUBZILKMSA-N Arg-Pro-Asp Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CC(O)=O)C(O)=O OVQJAKFLFTZDNC-GUBZILKMSA-N 0.000 description 2
- 108010051330 Arg-Pro-Gly-Pro Proteins 0.000 description 2
- YCYXHLZRUSJITQ-SRVKXCTJSA-N Arg-Pro-Pro Chemical compound NC(=N)NCCC[C@H](N)C(=O)N1CCC[C@H]1C(=O)N1[C@H](C(O)=O)CCC1 YCYXHLZRUSJITQ-SRVKXCTJSA-N 0.000 description 2
- VUGWHBXPMAHEGZ-SRVKXCTJSA-N Arg-Pro-Val Chemical compound CC(C)[C@@H](C(O)=O)NC(=O)[C@@H]1CCCN1C(=O)[C@@H](N)CCCN=C(N)N VUGWHBXPMAHEGZ-SRVKXCTJSA-N 0.000 description 2
- FRBAHXABMQXSJQ-FXQIFTODSA-N Arg-Ser-Ser Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CO)C(=O)N[C@@H](CO)C(O)=O FRBAHXABMQXSJQ-FXQIFTODSA-N 0.000 description 2
- QLSRIZIDQXDQHK-RCWTZXSCSA-N Arg-Val-Thr Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O QLSRIZIDQXDQHK-RCWTZXSCSA-N 0.000 description 2
- WPOLSNAQGVHROR-GUBZILKMSA-N Asn-Gln-Leu Chemical compound CC(C)C[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CC(=O)N)N WPOLSNAQGVHROR-GUBZILKMSA-N 0.000 description 2
- VITDJIPIJZAVGC-VEVYYDQMSA-N Asn-Met-Thr Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CCSC)C(=O)N[C@@H]([C@@H](C)O)C(O)=O VITDJIPIJZAVGC-VEVYYDQMSA-N 0.000 description 2
- YXVAESUIQFDBHN-SRVKXCTJSA-N Asn-Phe-Ser Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CO)C(O)=O YXVAESUIQFDBHN-SRVKXCTJSA-N 0.000 description 2
- RTXQQDVBACBSCW-CFMVVWHZSA-N Asp-Ile-Tyr Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O RTXQQDVBACBSCW-CFMVVWHZSA-N 0.000 description 2
- UZFHNLYQWMGUHU-DCAQKATOSA-N Asp-Lys-Arg Chemical compound OC(=O)C[C@H](N)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O UZFHNLYQWMGUHU-DCAQKATOSA-N 0.000 description 2
- GCACQYDBDHRVGE-LKXGYXEUSA-N Asp-Thr-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@H]([C@H](O)C)NC(=O)[C@@H](N)CC(O)=O GCACQYDBDHRVGE-LKXGYXEUSA-N 0.000 description 2
- JDDYEZGPYBBPBN-JRQIVUDYSA-N Asp-Thr-Tyr Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O JDDYEZGPYBBPBN-JRQIVUDYSA-N 0.000 description 2
- WOKXEQLPBLLWHC-IHRRRGAJSA-N Asp-Tyr-Met Chemical compound CSCC[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)[C@@H](N)CC(O)=O)CC1=CC=C(O)C=C1 WOKXEQLPBLLWHC-IHRRRGAJSA-N 0.000 description 2
- HTSSXFASOUSJQG-IHPCNDPISA-N Asp-Tyr-Trp Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(O)=O HTSSXFASOUSJQG-IHPCNDPISA-N 0.000 description 2
- GIKOVDMXBAFXDF-NHCYSSNCSA-N Asp-Val-Leu Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CC(C)C)C(O)=O GIKOVDMXBAFXDF-NHCYSSNCSA-N 0.000 description 2
- IJGRMHOSHXDMSA-UHFFFAOYSA-N Atomic nitrogen Chemical compound N#N IJGRMHOSHXDMSA-UHFFFAOYSA-N 0.000 description 2
- 208000004736 B-Cell Leukemia Diseases 0.000 description 2
- 241000283690 Bos taurus Species 0.000 description 2
- 238000011523 CAR-T cell immunotherapy Methods 0.000 description 2
- 102100037904 CD9 antigen Human genes 0.000 description 2
- 108010021064 CTLA-4 Antigen Proteins 0.000 description 2
- 229940045513 CTLA4 antagonist Drugs 0.000 description 2
- 241000282836 Camelus dromedarius Species 0.000 description 2
- 108090000566 Caspase-9 Proteins 0.000 description 2
- 206010009900 Colitis ulcerative Diseases 0.000 description 2
- 208000011231 Crohn disease Diseases 0.000 description 2
- KABHAOSDMIYXTR-GUBZILKMSA-N Cys-Glu-Leu Chemical compound CC(C)C[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)O)NC(=O)[C@H](CS)N KABHAOSDMIYXTR-GUBZILKMSA-N 0.000 description 2
- OZHXXYOHPLLLMI-CIUDSAMLSA-N Cys-Lys-Ala Chemical compound [H]N[C@@H](CS)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](C)C(O)=O OZHXXYOHPLLLMI-CIUDSAMLSA-N 0.000 description 2
- 108010015742 Cytochrome P-450 Enzyme System Proteins 0.000 description 2
- 102000003849 Cytochrome P450 Human genes 0.000 description 2
- 102100039498 Cytotoxic T-lymphocyte protein 4 Human genes 0.000 description 2
- 241000702421 Dependoparvovirus Species 0.000 description 2
- 201000004624 Dermatitis Diseases 0.000 description 2
- 241000214054 Equine rhinitis A virus Species 0.000 description 2
- 241000283073 Equus caballus Species 0.000 description 2
- 241000282326 Felis catus Species 0.000 description 2
- TWHDOEYLXXQYOZ-FXQIFTODSA-N Gln-Asn-Gln Chemical compound C(CC(=O)N)[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H](CCC(=O)N)C(=O)O)N TWHDOEYLXXQYOZ-FXQIFTODSA-N 0.000 description 2
- NVEASDQHBRZPSU-BQBZGAKWSA-N Gln-Gln-Gly Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCC(N)=O)C(=O)NCC(O)=O NVEASDQHBRZPSU-BQBZGAKWSA-N 0.000 description 2
- MADFVRSKEIEZHZ-DCAQKATOSA-N Gln-Gln-Lys Chemical compound C(CCN)C[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CCC(=O)N)N MADFVRSKEIEZHZ-DCAQKATOSA-N 0.000 description 2
- CLPQUWHBWXFJOX-BQBZGAKWSA-N Gln-Gly-Gln Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)NCC(=O)N[C@@H](CCC(N)=O)C(O)=O CLPQUWHBWXFJOX-BQBZGAKWSA-N 0.000 description 2
- UTKICHUQEQBDGC-ACZMJKKPSA-N Glu-Ala-Cys Chemical compound C[C@@H](C(=O)N[C@@H](CS)C(=O)O)NC(=O)[C@H](CCC(=O)O)N UTKICHUQEQBDGC-ACZMJKKPSA-N 0.000 description 2
- BUZMZDDKFCSKOT-CIUDSAMLSA-N Glu-Glu-Glu Chemical compound OC(=O)CC[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCC(O)=O)C(O)=O BUZMZDDKFCSKOT-CIUDSAMLSA-N 0.000 description 2
- BUAKRRKDHSSIKK-IHRRRGAJSA-N Glu-Glu-Tyr Chemical compound OC(=O)CC[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 BUAKRRKDHSSIKK-IHRRRGAJSA-N 0.000 description 2
- OAGVHWYIBZMWLA-YFKPBYRVSA-N Glu-Gly-Gly Chemical compound OC(=O)CC[C@H](N)C(=O)NCC(=O)NCC(O)=O OAGVHWYIBZMWLA-YFKPBYRVSA-N 0.000 description 2
- VMKCPNBBPGGQBJ-GUBZILKMSA-N Glu-Leu-Asn Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)O)NC(=O)[C@H](CCC(=O)O)N VMKCPNBBPGGQBJ-GUBZILKMSA-N 0.000 description 2
- DNPCBMNFQVTHMA-DCAQKATOSA-N Glu-Leu-Gln Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(N)=O)C(O)=O DNPCBMNFQVTHMA-DCAQKATOSA-N 0.000 description 2
- QMOSCLNJVKSHHU-YUMQZZPRSA-N Glu-Met-Gly Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCSC)C(=O)NCC(O)=O QMOSCLNJVKSHHU-YUMQZZPRSA-N 0.000 description 2
- HHSKZJZWQFPSKN-AVGNSLFASA-N Glu-Tyr-Asp Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(O)=O)C(O)=O HHSKZJZWQFPSKN-AVGNSLFASA-N 0.000 description 2
- QSDKBRMVXSWAQE-BFHQHQDPSA-N Gly-Ala-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)[C@H](C)NC(=O)CN QSDKBRMVXSWAQE-BFHQHQDPSA-N 0.000 description 2
- CCQOOWAONKGYKQ-BYPYZUCNSA-N Gly-Gly-Ala Chemical compound OC(=O)[C@H](C)NC(=O)CNC(=O)CN CCQOOWAONKGYKQ-BYPYZUCNSA-N 0.000 description 2
- QITBQGJOXQYMOA-ZETCQYMHSA-N Gly-Gly-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)CNC(=O)CN QITBQGJOXQYMOA-ZETCQYMHSA-N 0.000 description 2
- YWAQATDNEKZFFK-BYPYZUCNSA-N Gly-Gly-Ser Chemical compound NCC(=O)NCC(=O)N[C@@H](CO)C(O)=O YWAQATDNEKZFFK-BYPYZUCNSA-N 0.000 description 2
- ULZCYBYDTUMHNF-IUCAKERBSA-N Gly-Leu-Glu Chemical compound NCC(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(O)=O)C(O)=O ULZCYBYDTUMHNF-IUCAKERBSA-N 0.000 description 2
- AFWYPMDMDYCKMD-KBPBESRZSA-N Gly-Leu-Tyr Chemical compound NCC(=O)N[C@@H](CC(C)C)C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 AFWYPMDMDYCKMD-KBPBESRZSA-N 0.000 description 2
- VBOBNHSVQKKTOT-YUMQZZPRSA-N Gly-Lys-Ala Chemical compound [H]NCC(=O)N[C@@H](CCCCN)C(=O)N[C@@H](C)C(O)=O VBOBNHSVQKKTOT-YUMQZZPRSA-N 0.000 description 2
- WDEHMRNSGHVNOH-VHSXEESVSA-N Gly-Lys-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CCCCN)NC(=O)CN)C(=O)O WDEHMRNSGHVNOH-VHSXEESVSA-N 0.000 description 2
- GLACUWHUYFBSPJ-FJXKBIBVSA-N Gly-Pro-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)[C@@H]1CCCN1C(=O)CN GLACUWHUYFBSPJ-FJXKBIBVSA-N 0.000 description 2
- FGPLUIQCSKGLTI-WDSKDSINSA-N Gly-Ser-Glu Chemical compound NCC(=O)N[C@@H](CO)C(=O)N[C@H](C(O)=O)CCC(O)=O FGPLUIQCSKGLTI-WDSKDSINSA-N 0.000 description 2
- XHVONGZZVUUORG-WEDXCCLWSA-N Gly-Thr-Lys Chemical compound NCC(=O)N[C@@H]([C@H](O)C)C(=O)N[C@H](C(O)=O)CCCCN XHVONGZZVUUORG-WEDXCCLWSA-N 0.000 description 2
- BAYQNCWLXIDLHX-ONGXEEELSA-N Gly-Val-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](C(C)C)NC(=O)CN BAYQNCWLXIDLHX-ONGXEEELSA-N 0.000 description 2
- YGHSQRJSHKYUJY-SCZZXKLOSA-N Gly-Val-Pro Chemical compound CC(C)[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)CN YGHSQRJSHKYUJY-SCZZXKLOSA-N 0.000 description 2
- 102000003886 Glycoproteins Human genes 0.000 description 2
- 108090000288 Glycoproteins Proteins 0.000 description 2
- 102000010956 Glypican Human genes 0.000 description 2
- 108050001154 Glypican Proteins 0.000 description 2
- 108050007237 Glypican-3 Proteins 0.000 description 2
- BIAKMWKJMQLZOJ-ZKWXMUAHSA-N His-Ala-Ala Chemical compound C[C@H](NC(=O)[C@H](C)NC(=O)[C@@H](N)Cc1cnc[nH]1)C(O)=O BIAKMWKJMQLZOJ-ZKWXMUAHSA-N 0.000 description 2
- 101000738354 Homo sapiens CD9 antigen Proteins 0.000 description 2
- 101000777628 Homo sapiens Leukocyte antigen CD37 Proteins 0.000 description 2
- 101000917858 Homo sapiens Low affinity immunoglobulin gamma Fc region receptor III-A Proteins 0.000 description 2
- 101000917839 Homo sapiens Low affinity immunoglobulin gamma Fc region receptor III-B Proteins 0.000 description 2
- HDOYNXLPTRQLAD-JBDRJPRFSA-N Ile-Ala-Ser Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](C)C(=O)N[C@@H](CO)C(=O)O)N HDOYNXLPTRQLAD-JBDRJPRFSA-N 0.000 description 2
- JTBFQNHKNRZJDS-SYWGBEHUSA-N Ile-Trp-Ala Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC1=CNC2=CC=CC=C21)C(=O)N[C@@H](C)C(=O)O)N JTBFQNHKNRZJDS-SYWGBEHUSA-N 0.000 description 2
- 206010061218 Inflammation Diseases 0.000 description 2
- 208000022559 Inflammatory bowel disease Diseases 0.000 description 2
- 102000013462 Interleukin-12 Human genes 0.000 description 2
- 108010065805 Interleukin-12 Proteins 0.000 description 2
- 108010002616 Interleukin-5 Proteins 0.000 description 2
- 108010002586 Interleukin-7 Proteins 0.000 description 2
- 102000000704 Interleukin-7 Human genes 0.000 description 2
- SITWEMZOJNKJCH-UHFFFAOYSA-N L-alanine-L-arginine Natural products CC(N)C(=O)NC(C(O)=O)CCCNC(N)=N SITWEMZOJNKJCH-UHFFFAOYSA-N 0.000 description 2
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 2
- KZSNJWFQEVHDMF-BYPYZUCNSA-N L-valine Chemical compound CC(C)[C@H](N)C(O)=O KZSNJWFQEVHDMF-BYPYZUCNSA-N 0.000 description 2
- 241000880493 Leptailurus serval Species 0.000 description 2
- MYGQXVYRZMKRDB-SRVKXCTJSA-N Leu-Asp-Lys Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@H](C(O)=O)CCCCN MYGQXVYRZMKRDB-SRVKXCTJSA-N 0.000 description 2
- OYQUOLRTJHWVSQ-SRVKXCTJSA-N Leu-His-Ser Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CO)C(O)=O OYQUOLRTJHWVSQ-SRVKXCTJSA-N 0.000 description 2
- ZGUMORRUBUCXEH-AVGNSLFASA-N Leu-Lys-Gln Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCC(N)=O)C(O)=O ZGUMORRUBUCXEH-AVGNSLFASA-N 0.000 description 2
- DPURXCQCHSQPAN-AVGNSLFASA-N Leu-Pro-Pro Chemical compound CC(C)C[C@H](N)C(=O)N1CCC[C@H]1C(=O)N1[C@H](C(O)=O)CCC1 DPURXCQCHSQPAN-AVGNSLFASA-N 0.000 description 2
- IRMLZWSRWSGTOP-CIUDSAMLSA-N Leu-Ser-Ala Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CO)C(=O)N[C@@H](C)C(O)=O IRMLZWSRWSGTOP-CIUDSAMLSA-N 0.000 description 2
- PPGBXYKMUMHFBF-KATARQTJSA-N Leu-Ser-Thr Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(=O)N[C@@H]([C@@H](C)O)C(O)=O PPGBXYKMUMHFBF-KATARQTJSA-N 0.000 description 2
- ILDSIMPXNFWKLH-KATARQTJSA-N Leu-Thr-Ser Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CO)C(O)=O ILDSIMPXNFWKLH-KATARQTJSA-N 0.000 description 2
- ARNIBBOXIAWUOP-MGHWNKPDSA-N Leu-Tyr-Ile Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O ARNIBBOXIAWUOP-MGHWNKPDSA-N 0.000 description 2
- 102100031586 Leukocyte antigen CD37 Human genes 0.000 description 2
- 206010058467 Lung neoplasm malignant Diseases 0.000 description 2
- LMVOVCYVZBBWQB-SRVKXCTJSA-N Lys-Asp-Lys Chemical compound NCCCC[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@H](C(O)=O)CCCCN LMVOVCYVZBBWQB-SRVKXCTJSA-N 0.000 description 2
- LCMWVZLBCUVDAZ-IUCAKERBSA-N Lys-Gly-Glu Chemical compound [NH3+]CCCC[C@H]([NH3+])C(=O)NCC(=O)N[C@H](C([O-])=O)CCC([O-])=O LCMWVZLBCUVDAZ-IUCAKERBSA-N 0.000 description 2
- UETQMSASAVBGJY-QWRGUYRKSA-N Lys-Gly-His Chemical compound NCCCC[C@H](N)C(=O)NCC(=O)N[C@H](C(O)=O)CC1=CNC=N1 UETQMSASAVBGJY-QWRGUYRKSA-N 0.000 description 2
- GQFDWEDHOQRNLC-QWRGUYRKSA-N Lys-Gly-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)CNC(=O)[C@@H](N)CCCCN GQFDWEDHOQRNLC-QWRGUYRKSA-N 0.000 description 2
- HVAUKHLDSDDROB-KKUMJFAQSA-N Lys-Lys-Leu Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(C)C)C(O)=O HVAUKHLDSDDROB-KKUMJFAQSA-N 0.000 description 2
- AZOFEHCPMBRNFD-BZSNNMDCSA-N Lys-Phe-Lys Chemical compound NCCCC[C@H](N)C(=O)N[C@H](C(=O)N[C@@H](CCCCN)C(O)=O)CC1=CC=CC=C1 AZOFEHCPMBRNFD-BZSNNMDCSA-N 0.000 description 2
- ONGCSGVHCSAATF-CIUDSAMLSA-N Met-Ala-Glu Chemical compound CSCC[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@H](C(O)=O)CCC(O)=O ONGCSGVHCSAATF-CIUDSAMLSA-N 0.000 description 2
- QEVRUYFHWJJUHZ-DCAQKATOSA-N Met-Ala-Leu Chemical compound CSCC[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@H](C(O)=O)CC(C)C QEVRUYFHWJJUHZ-DCAQKATOSA-N 0.000 description 2
- YLLWCSDBVGZLOW-CIUDSAMLSA-N Met-Gln-Ala Chemical compound [H]N[C@@H](CCSC)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](C)C(O)=O YLLWCSDBVGZLOW-CIUDSAMLSA-N 0.000 description 2
- 206010029260 Neuroblastoma Diseases 0.000 description 2
- 102000015636 Oligopeptides Human genes 0.000 description 2
- 108010038807 Oligopeptides Proteins 0.000 description 2
- BJEYSVHMGIJORT-NHCYSSNCSA-N Phe-Ala-Ala Chemical compound OC(=O)[C@H](C)NC(=O)[C@H](C)NC(=O)[C@@H](N)CC1=CC=CC=C1 BJEYSVHMGIJORT-NHCYSSNCSA-N 0.000 description 2
- MJAYDXWQQUOURZ-JYJNAYRXSA-N Phe-Lys-Gln Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCC(N)=O)C(O)=O MJAYDXWQQUOURZ-JYJNAYRXSA-N 0.000 description 2
- 102100028251 Phosphoglycerate kinase 1 Human genes 0.000 description 2
- 101710139464 Phosphoglycerate kinase 1 Proteins 0.000 description 2
- 102100037935 Polyubiquitin-C Human genes 0.000 description 2
- 208000009052 Precursor T-Cell Lymphoblastic Leukemia-Lymphoma Diseases 0.000 description 2
- 208000017414 Precursor T-cell acute lymphoblastic leukemia Diseases 0.000 description 2
- DZZCICYRSZASNF-FXQIFTODSA-N Pro-Ala-Ala Chemical compound OC(=O)[C@H](C)NC(=O)[C@H](C)NC(=O)[C@@H]1CCCN1 DZZCICYRSZASNF-FXQIFTODSA-N 0.000 description 2
- CGBYDGAJHSOGFQ-LPEHRKFASA-N Pro-Ala-Pro Chemical compound C[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@@H]2CCCN2 CGBYDGAJHSOGFQ-LPEHRKFASA-N 0.000 description 2
- LNLNHXIQPGKRJQ-SRVKXCTJSA-N Pro-Arg-Arg Chemical compound NC(N)=NCCC[C@@H](C(O)=O)NC(=O)[C@H](CCCN=C(N)N)NC(=O)[C@@H]1CCCN1 LNLNHXIQPGKRJQ-SRVKXCTJSA-N 0.000 description 2
- SSSFPISOZOLQNP-GUBZILKMSA-N Pro-Arg-Asp Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(O)=O)C(O)=O SSSFPISOZOLQNP-GUBZILKMSA-N 0.000 description 2
- LSIWVWRUTKPXDS-DCAQKATOSA-N Pro-Gln-Arg Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O LSIWVWRUTKPXDS-DCAQKATOSA-N 0.000 description 2
- UPJGUQPLYWTISV-GUBZILKMSA-N Pro-Gln-Glu Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCC(O)=O)C(O)=O UPJGUQPLYWTISV-GUBZILKMSA-N 0.000 description 2
- PTLOFJZJADCNCD-DCAQKATOSA-N Pro-Glu-Met Chemical compound CSCC[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)O)NC(=O)[C@@H]1CCCN1 PTLOFJZJADCNCD-DCAQKATOSA-N 0.000 description 2
- RMODQFBNDDENCP-IHRRRGAJSA-N Pro-Lys-Leu Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(C)C)C(O)=O RMODQFBNDDENCP-IHRRRGAJSA-N 0.000 description 2
- DSGSTPRKNYHGCL-JYJNAYRXSA-N Pro-Phe-Met Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CCSC)C(O)=O DSGSTPRKNYHGCL-JYJNAYRXSA-N 0.000 description 2
- JLMZKEQFMVORMA-SRVKXCTJSA-N Pro-Pro-Arg Chemical compound NC(N)=NCCC[C@@H](C(O)=O)NC(=O)[C@@H]1CCCN1C(=O)[C@H]1NCCC1 JLMZKEQFMVORMA-SRVKXCTJSA-N 0.000 description 2
- CHYAYDLYYIJCKY-OSUNSFLBSA-N Pro-Thr-Ile Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O CHYAYDLYYIJCKY-OSUNSFLBSA-N 0.000 description 2
- AIOWVDNPESPXRB-YTWAJWBKSA-N Pro-Thr-Pro Chemical compound C[C@H]([C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@@H]2CCCN2)O AIOWVDNPESPXRB-YTWAJWBKSA-N 0.000 description 2
- QKWYXRPICJEQAJ-KJEVXHAQSA-N Pro-Tyr-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CC1=CC=C(C=C1)O)NC(=O)[C@@H]2CCCN2)O QKWYXRPICJEQAJ-KJEVXHAQSA-N 0.000 description 2
- RJKFOVLPORLFTN-LEKSSAKUSA-N Progesterone Chemical compound C1CC2=CC(=O)CC[C@]2(C)[C@@H]2[C@@H]1[C@@H]1CC[C@H](C(=O)C)[C@@]1(C)CC2 RJKFOVLPORLFTN-LEKSSAKUSA-N 0.000 description 2
- 206010060862 Prostate cancer Diseases 0.000 description 2
- 208000000236 Prostatic Neoplasms Diseases 0.000 description 2
- 201000004681 Psoriasis Diseases 0.000 description 2
- 208000007660 Residual Neoplasm Diseases 0.000 description 2
- LVVBAKCGXXUHFO-ZLUOBGJFSA-N Ser-Ala-Asp Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](C)C(=O)N[C@@H](CC(O)=O)C(O)=O LVVBAKCGXXUHFO-ZLUOBGJFSA-N 0.000 description 2
- QGMLKFGTGXWAHF-IHRRRGAJSA-N Ser-Arg-Phe Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O QGMLKFGTGXWAHF-IHRRRGAJSA-N 0.000 description 2
- HVKMTOIAYDOJPL-NRPADANISA-N Ser-Gln-Val Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](C(C)C)C(O)=O HVKMTOIAYDOJPL-NRPADANISA-N 0.000 description 2
- SFTZWNJFZYOLBD-ZDLURKLDSA-N Ser-Gly-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)CNC(=O)[C@@H](N)CO SFTZWNJFZYOLBD-ZDLURKLDSA-N 0.000 description 2
- XXXAXOWMBOKTRN-XPUUQOCRSA-N Ser-Gly-Val Chemical compound [H]N[C@@H](CO)C(=O)NCC(=O)N[C@@H](C(C)C)C(O)=O XXXAXOWMBOKTRN-XPUUQOCRSA-N 0.000 description 2
- QYSFWUIXDFJUDW-DCAQKATOSA-N Ser-Leu-Arg Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O QYSFWUIXDFJUDW-DCAQKATOSA-N 0.000 description 2
- YUJLIIRMIAGMCQ-CIUDSAMLSA-N Ser-Leu-Ser Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(O)=O YUJLIIRMIAGMCQ-CIUDSAMLSA-N 0.000 description 2
- JURQXQBJKUHGJS-UHFFFAOYSA-N Ser-Ser-Ser-Ser Chemical compound OCC(N)C(=O)NC(CO)C(=O)NC(CO)C(=O)NC(CO)C(O)=O JURQXQBJKUHGJS-UHFFFAOYSA-N 0.000 description 2
- PYTKULIABVRXSC-BWBBJGPYSA-N Ser-Ser-Thr Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CO)C(=O)N[C@@H]([C@@H](C)O)C(O)=O PYTKULIABVRXSC-BWBBJGPYSA-N 0.000 description 2
- VGQVAVQWKJLIRM-FXQIFTODSA-N Ser-Ser-Val Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(O)=O VGQVAVQWKJLIRM-FXQIFTODSA-N 0.000 description 2
- XJDMUQCLVSCRSJ-VZFHVOOUSA-N Ser-Thr-Ala Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C)C(O)=O XJDMUQCLVSCRSJ-VZFHVOOUSA-N 0.000 description 2
- SIEBDTCABMZCLF-XGEHTFHBSA-N Ser-Val-Thr Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O SIEBDTCABMZCLF-XGEHTFHBSA-N 0.000 description 2
- 208000021386 Sjogren Syndrome Diseases 0.000 description 2
- 201000009594 Systemic Scleroderma Diseases 0.000 description 2
- 206010042953 Systemic sclerosis Diseases 0.000 description 2
- 230000006052 T cell proliferation Effects 0.000 description 2
- 208000029052 T-cell acute lymphoblastic leukemia Diseases 0.000 description 2
- 239000004098 Tetracycline Substances 0.000 description 2
- FQPDRTDDEZXCEC-SVSWQMSJSA-N Thr-Ile-Ser Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CO)C(O)=O FQPDRTDDEZXCEC-SVSWQMSJSA-N 0.000 description 2
- SPVHQURZJCUDQC-VOAKCMCISA-N Thr-Lys-Leu Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(C)C)C(O)=O SPVHQURZJCUDQC-VOAKCMCISA-N 0.000 description 2
- STUAPCLEDMKXKL-LKXGYXEUSA-N Thr-Ser-Asn Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(N)=O)C(O)=O STUAPCLEDMKXKL-LKXGYXEUSA-N 0.000 description 2
- WPSKTVVMQCXPRO-BWBBJGPYSA-N Thr-Ser-Ser Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CO)C(O)=O WPSKTVVMQCXPRO-BWBBJGPYSA-N 0.000 description 2
- COYHRQWNJDJCNA-NUJDXYNKSA-N Thr-Thr-Thr Chemical compound C[C@@H](O)[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O COYHRQWNJDJCNA-NUJDXYNKSA-N 0.000 description 2
- YOPQYBJJNSIQGZ-JNPHEJMOSA-N Thr-Tyr-Tyr Chemical compound C([C@H](NC(=O)[C@@H](N)[C@H](O)C)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(O)=O)C1=CC=C(O)C=C1 YOPQYBJJNSIQGZ-JNPHEJMOSA-N 0.000 description 2
- 102000006601 Thymidine Kinase Human genes 0.000 description 2
- 108020004440 Thymidine kinase Proteins 0.000 description 2
- 108700019146 Transgenes Proteins 0.000 description 2
- 102100040247 Tumor necrosis factor Human genes 0.000 description 2
- OOEUVMFKKZYSRX-LEWSCRJBSA-N Tyr-Ala-Pro Chemical compound C[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CC2=CC=C(C=C2)O)N OOEUVMFKKZYSRX-LEWSCRJBSA-N 0.000 description 2
- PEVVXUGSAKEPEN-AVGNSLFASA-N Tyr-Asn-Glu Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CCC(O)=O)C(O)=O PEVVXUGSAKEPEN-AVGNSLFASA-N 0.000 description 2
- RYSNTWVRSLCAJZ-RYUDHWBXSA-N Tyr-Gln-Gly Chemical compound OC(=O)CNC(=O)[C@H](CCC(N)=O)NC(=O)[C@@H](N)CC1=CC=C(O)C=C1 RYSNTWVRSLCAJZ-RYUDHWBXSA-N 0.000 description 2
- CKHQKYHIZCRTAP-SOUVJXGZSA-N Tyr-Gln-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CC2=CC=C(C=C2)O)N)C(=O)O CKHQKYHIZCRTAP-SOUVJXGZSA-N 0.000 description 2
- HIINQLBHPIQYHN-JTQLQIEISA-N Tyr-Gly-Gly Chemical compound OC(=O)CNC(=O)CNC(=O)[C@@H](N)CC1=CC=C(O)C=C1 HIINQLBHPIQYHN-JTQLQIEISA-N 0.000 description 2
- ANHVRCNNGJMJNG-BZSNNMDCSA-N Tyr-Tyr-Cys Chemical compound C1=CC(=CC=C1C[C@@H](C(=O)N[C@@H](CC2=CC=C(C=C2)O)C(=O)N[C@@H](CS)C(=O)O)N)O ANHVRCNNGJMJNG-BZSNNMDCSA-N 0.000 description 2
- 108010056354 Ubiquitin C Proteins 0.000 description 2
- 201000006704 Ulcerative Colitis Diseases 0.000 description 2
- ZLFHAAGHGQBQQN-AEJSXWLSSA-N Val-Ala-Pro Chemical compound C[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](C(C)C)N ZLFHAAGHGQBQQN-AEJSXWLSSA-N 0.000 description 2
- ZLFHAAGHGQBQQN-GUBZILKMSA-N Val-Ala-Pro Natural products CC(C)[C@H](N)C(=O)N[C@@H](C)C(=O)N1CCC[C@H]1C(O)=O ZLFHAAGHGQBQQN-GUBZILKMSA-N 0.000 description 2
- VFOHXOLPLACADK-GVXVVHGQSA-N Val-Gln-Leu Chemical compound CC(C)C[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](C(C)C)N VFOHXOLPLACADK-GVXVVHGQSA-N 0.000 description 2
- ZRSZTKTVPNSUNA-IHRRRGAJSA-N Val-Lys-Leu Chemical compound CC(C)C[C@H](NC(=O)[C@H](CCCCN)NC(=O)[C@@H](N)C(C)C)C(O)=O ZRSZTKTVPNSUNA-IHRRRGAJSA-N 0.000 description 2
- KZSNJWFQEVHDMF-UHFFFAOYSA-N Valine Natural products CC(C)C(N)C(O)=O KZSNJWFQEVHDMF-UHFFFAOYSA-N 0.000 description 2
- 206010047115 Vasculitis Diseases 0.000 description 2
- 108010017070 Zinc Finger Nucleases Proteins 0.000 description 2
- 230000009471 action Effects 0.000 description 2
- 108010050025 alpha-glutamyltryptophan Proteins 0.000 description 2
- 125000000539 amino acid group Chemical group 0.000 description 2
- 230000001093 anti-cancer Effects 0.000 description 2
- 230000030741 antigen processing and presentation Effects 0.000 description 2
- 230000000890 antigenic effect Effects 0.000 description 2
- 108010069205 aspartyl-phenylalanine Proteins 0.000 description 2
- 230000008901 benefit Effects 0.000 description 2
- 230000004071 biological effect Effects 0.000 description 2
- 230000017531 blood circulation Effects 0.000 description 2
- 210000001772 blood platelet Anatomy 0.000 description 2
- 238000010322 bone marrow transplantation Methods 0.000 description 2
- 230000004663 cell proliferation Effects 0.000 description 2
- 208000024207 chronic leukemia Diseases 0.000 description 2
- 238000010367 cloning Methods 0.000 description 2
- 230000006690 co-activation Effects 0.000 description 2
- 230000001086 cytosolic effect Effects 0.000 description 2
- 229940127089 cytotoxic agent Drugs 0.000 description 2
- 230000006378 damage Effects 0.000 description 2
- 230000003247 decreasing effect Effects 0.000 description 2
- 230000001419 dependent effect Effects 0.000 description 2
- 238000011161 development Methods 0.000 description 2
- 239000003814 drug Substances 0.000 description 2
- 230000009977 dual effect Effects 0.000 description 2
- 210000003162 effector t lymphocyte Anatomy 0.000 description 2
- 210000001671 embryonic stem cell Anatomy 0.000 description 2
- 238000013401 experimental design Methods 0.000 description 2
- 108010006664 gamma-glutamyl-glycyl-glycine Proteins 0.000 description 2
- 108010050475 glycyl-leucyl-tyrosine Proteins 0.000 description 2
- 239000001963 growth medium Substances 0.000 description 2
- 230000003394 haemopoietic effect Effects 0.000 description 2
- 210000005260 human cell Anatomy 0.000 description 2
- 230000002209 hydrophobic effect Effects 0.000 description 2
- 125000001165 hydrophobic group Chemical group 0.000 description 2
- 210000003297 immature b lymphocyte Anatomy 0.000 description 2
- 230000001976 improved effect Effects 0.000 description 2
- 230000005917 in vivo anti-tumor Effects 0.000 description 2
- 238000010832 independent-sample T-test Methods 0.000 description 2
- 208000015181 infectious disease Diseases 0.000 description 2
- 230000004054 inflammatory process Effects 0.000 description 2
- 230000002401 inhibitory effect Effects 0.000 description 2
- 230000005764 inhibitory process Effects 0.000 description 2
- 210000003734 kidney Anatomy 0.000 description 2
- 231100000518 lethal Toxicity 0.000 description 2
- 230000001665 lethal effect Effects 0.000 description 2
- 108010034529 leucyl-lysine Proteins 0.000 description 2
- 239000002502 liposome Substances 0.000 description 2
- 230000005923 long-lasting effect Effects 0.000 description 2
- 230000007774 longterm Effects 0.000 description 2
- 201000005202 lung cancer Diseases 0.000 description 2
- 208000020816 lung neoplasm Diseases 0.000 description 2
- 108010064235 lysylglycine Proteins 0.000 description 2
- 230000002101 lytic effect Effects 0.000 description 2
- 238000012423 maintenance Methods 0.000 description 2
- 210000001806 memory b lymphocyte Anatomy 0.000 description 2
- 108010005942 methionylglycine Proteins 0.000 description 2
- 230000004048 modification Effects 0.000 description 2
- 238000012986 modification Methods 0.000 description 2
- 210000001616 monocyte Anatomy 0.000 description 2
- 238000010172 mouse model Methods 0.000 description 2
- 201000006417 multiple sclerosis Diseases 0.000 description 2
- 230000002071 myeloproliferative effect Effects 0.000 description 2
- 230000002018 overexpression Effects 0.000 description 2
- 108010026466 polyproline Proteins 0.000 description 2
- 230000002265 prevention Effects 0.000 description 2
- 230000001737 promoting effect Effects 0.000 description 2
- 230000006337 proteolytic cleavage Effects 0.000 description 2
- 206010039073 rheumatoid arthritis Diseases 0.000 description 2
- 230000028327 secretion Effects 0.000 description 2
- 239000012679 serum free medium Substances 0.000 description 2
- 150000003384 small molecules Chemical class 0.000 description 2
- DAEPDZWVDSPTHF-UHFFFAOYSA-M sodium pyruvate Chemical compound [Na+].CC(=O)C([O-])=O DAEPDZWVDSPTHF-UHFFFAOYSA-M 0.000 description 2
- 241000894007 species Species 0.000 description 2
- 230000000638 stimulation Effects 0.000 description 2
- 239000000126 substance Substances 0.000 description 2
- 238000001356 surgical procedure Methods 0.000 description 2
- 201000000596 systemic lupus erythematosus Diseases 0.000 description 2
- 229930101283 tetracycline Natural products 0.000 description 2
- 229960002180 tetracycline Drugs 0.000 description 2
- 235000019364 tetracycline Nutrition 0.000 description 2
- 150000003522 tetracyclines Chemical class 0.000 description 2
- 231100000331 toxic Toxicity 0.000 description 2
- 230000002588 toxic effect Effects 0.000 description 2
- 230000001052 transient effect Effects 0.000 description 2
- 230000032258 transport Effects 0.000 description 2
- 229940121358 tyrosine kinase inhibitor Drugs 0.000 description 2
- 239000005483 tyrosine kinase inhibitor Substances 0.000 description 2
- 150000004917 tyrosine kinase inhibitor derivatives Chemical class 0.000 description 2
- 241000701161 unidentified adenovirus Species 0.000 description 2
- 239000004474 valine Substances 0.000 description 2
- 238000005406 washing Methods 0.000 description 2
- DIGQNXIGRZPYDK-WKSCXVIASA-N (2R)-6-amino-2-[[2-[[(2S)-2-[[2-[[(2R)-2-[[(2S)-2-[[(2R,3S)-2-[[2-[[(2S)-2-[[2-[[(2S)-2-[[(2S)-2-[[(2R)-2-[[(2S,3S)-2-[[(2R)-2-[[(2S)-2-[[(2S)-2-[[(2S)-2-[[2-[[(2S)-2-[[(2R)-2-[[2-[[2-[[2-[(2-amino-1-hydroxyethylidene)amino]-3-carboxy-1-hydroxypropylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1-hydroxyethylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1,3-dihydroxypropylidene]amino]-1-hydroxyethylidene]amino]-1-hydroxypropylidene]amino]-1,3-dihydroxypropylidene]amino]-1,3-dihydroxypropylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1,3-dihydroxybutylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1-hydroxypropylidene]amino]-1,3-dihydroxypropylidene]amino]-1-hydroxyethylidene]amino]-1,5-dihydroxy-5-iminopentylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1,3-dihydroxybutylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1,3-dihydroxypropylidene]amino]-1-hydroxyethylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1-hydroxyethylidene]amino]hexanoic acid Chemical compound C[C@@H]([C@@H](C(=N[C@@H](CS)C(=N[C@@H](C)C(=N[C@@H](CO)C(=NCC(=N[C@@H](CCC(=N)O)C(=NC(CS)C(=N[C@H]([C@H](C)O)C(=N[C@H](CS)C(=N[C@H](CO)C(=NCC(=N[C@H](CS)C(=NCC(=N[C@H](CCCCN)C(=O)O)O)O)O)O)O)O)O)O)O)O)O)O)O)N=C([C@H](CS)N=C([C@H](CO)N=C([C@H](CO)N=C([C@H](C)N=C(CN=C([C@H](CO)N=C([C@H](CS)N=C(CN=C(C(CS)N=C(C(CC(=O)O)N=C(CN)O)O)O)O)O)O)O)O)O)O)O)O DIGQNXIGRZPYDK-WKSCXVIASA-N 0.000 description 1
- JKMHFZQWWAIEOD-UHFFFAOYSA-N 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic acid Chemical compound OCC[NH+]1CCN(CCS([O-])(=O)=O)CC1 JKMHFZQWWAIEOD-UHFFFAOYSA-N 0.000 description 1
- QMOQBVOBWVNSNO-UHFFFAOYSA-N 2-[[2-[[2-[(2-azaniumylacetyl)amino]acetyl]amino]acetyl]amino]acetate Chemical compound NCC(=O)NCC(=O)NCC(=O)NCC(O)=O QMOQBVOBWVNSNO-UHFFFAOYSA-N 0.000 description 1
- QWTLUPDHBKBULE-UHFFFAOYSA-N 2-[[2-[[2-[[2-[[2-[[2-[[2-[[2-[[2-[(2-aminoacetyl)amino]acetyl]amino]acetyl]amino]acetyl]amino]acetyl]amino]acetyl]amino]acetyl]amino]acetyl]amino]acetyl]amino]acetic acid Chemical compound NCC(=O)NCC(=O)NCC(=O)NCC(=O)NCC(=O)NCC(=O)NCC(=O)NCC(=O)NCC(=O)NCC(O)=O QWTLUPDHBKBULE-UHFFFAOYSA-N 0.000 description 1
- 108010068327 4-hydroxyphenylpyruvate dioxygenase Proteins 0.000 description 1
- 108010027122 ADP-ribosyl Cyclase 1 Proteins 0.000 description 1
- 102000018667 ADP-ribosyl Cyclase 1 Human genes 0.000 description 1
- 102000007469 Actins Human genes 0.000 description 1
- 108010085238 Actins Proteins 0.000 description 1
- 208000036762 Acute promyelocytic leukaemia Diseases 0.000 description 1
- 208000026872 Addison Disease Diseases 0.000 description 1
- HHGYNJRJIINWAK-FXQIFTODSA-N Ala-Ala-Arg Chemical compound C[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@H](C(O)=O)CCCN=C(N)N HHGYNJRJIINWAK-FXQIFTODSA-N 0.000 description 1
- RLMISHABBKUNFO-WHFBIAKZSA-N Ala-Ala-Gly Chemical compound C[C@H](N)C(=O)N[C@@H](C)C(=O)NCC(O)=O RLMISHABBKUNFO-WHFBIAKZSA-N 0.000 description 1
- JBVSSSZFNTXJDX-YTLHQDLWSA-N Ala-Ala-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)[C@H](C)NC(=O)[C@H](C)N JBVSSSZFNTXJDX-YTLHQDLWSA-N 0.000 description 1
- UCIYCBSJBQGDGM-LPEHRKFASA-N Ala-Arg-Pro Chemical compound C[C@@H](C(=O)N[C@@H](CCCN=C(N)N)C(=O)N1CCC[C@@H]1C(=O)O)N UCIYCBSJBQGDGM-LPEHRKFASA-N 0.000 description 1
- LBJYAILUMSUTAM-ZLUOBGJFSA-N Ala-Asn-Asn Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CC(N)=O)C(O)=O LBJYAILUMSUTAM-ZLUOBGJFSA-N 0.000 description 1
- MBWYUTNBYSSUIQ-HERUPUMHSA-N Ala-Asn-Trp Chemical compound C[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H](CC1=CNC2=CC=CC=C21)C(=O)O)N MBWYUTNBYSSUIQ-HERUPUMHSA-N 0.000 description 1
- NHCPCLJZRSIDHS-ZLUOBGJFSA-N Ala-Asp-Ala Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](C)C(O)=O NHCPCLJZRSIDHS-ZLUOBGJFSA-N 0.000 description 1
- BTYTYHBSJKQBQA-GCJQMDKQSA-N Ala-Asp-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CC(=O)O)NC(=O)[C@H](C)N)O BTYTYHBSJKQBQA-GCJQMDKQSA-N 0.000 description 1
- WJRXVTCKASUIFF-FXQIFTODSA-N Ala-Cys-Arg Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CS)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O WJRXVTCKASUIFF-FXQIFTODSA-N 0.000 description 1
- DAEFQZCYZKRTLR-ZLUOBGJFSA-N Ala-Cys-Asp Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CS)C(=O)N[C@@H](CC(O)=O)C(O)=O DAEFQZCYZKRTLR-ZLUOBGJFSA-N 0.000 description 1
- FUSPCLTUKXQREV-ACZMJKKPSA-N Ala-Glu-Ala Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](C)C(O)=O FUSPCLTUKXQREV-ACZMJKKPSA-N 0.000 description 1
- HXNNRBHASOSVPG-GUBZILKMSA-N Ala-Glu-Leu Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(O)=O HXNNRBHASOSVPG-GUBZILKMSA-N 0.000 description 1
- VGPWRRFOPXVGOH-BYPYZUCNSA-N Ala-Gly-Gly Chemical compound C[C@H](N)C(=O)NCC(=O)NCC(O)=O VGPWRRFOPXVGOH-BYPYZUCNSA-N 0.000 description 1
- PCIFXPRIFWKWLK-YUMQZZPRSA-N Ala-Gly-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)CNC(=O)[C@H](C)N PCIFXPRIFWKWLK-YUMQZZPRSA-N 0.000 description 1
- OQWQTGBOFPJOIF-DLOVCJGASA-N Ala-Lys-His Chemical compound C[C@@H](C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)N OQWQTGBOFPJOIF-DLOVCJGASA-N 0.000 description 1
- DYXOFPBJBAHWFY-JBDRJPRFSA-N Ala-Ser-Ile Chemical compound CC[C@H](C)[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@H](C)N DYXOFPBJBAHWFY-JBDRJPRFSA-N 0.000 description 1
- HOVPGJUNRLMIOZ-CIUDSAMLSA-N Ala-Ser-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@H](C)N HOVPGJUNRLMIOZ-CIUDSAMLSA-N 0.000 description 1
- ARHJJAAWNWOACN-FXQIFTODSA-N Ala-Ser-Val Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(O)=O ARHJJAAWNWOACN-FXQIFTODSA-N 0.000 description 1
- IOFVWPYSRSCWHI-JXUBOQSCSA-N Ala-Thr-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H]([C@@H](C)O)NC(=O)[C@H](C)N IOFVWPYSRSCWHI-JXUBOQSCSA-N 0.000 description 1
- QDGMZAOSMNGBLP-MRFFXTKBSA-N Ala-Trp-Tyr Chemical compound C[C@@H](C(=O)N[C@@H](CC1=CNC2=CC=CC=C21)C(=O)N[C@@H](CC3=CC=C(C=C3)O)C(=O)O)N QDGMZAOSMNGBLP-MRFFXTKBSA-N 0.000 description 1
- BHFOJPDOQPWJRN-XDTLVQLUSA-N Ala-Tyr-Gln Chemical compound C[C@H](N)C(=O)N[C@@H](Cc1ccc(O)cc1)C(=O)N[C@@H](CCC(N)=O)C(O)=O BHFOJPDOQPWJRN-XDTLVQLUSA-N 0.000 description 1
- ZJLORAAXDAJLDC-CQDKDKBSSA-N Ala-Tyr-Leu Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(C)C)C(O)=O ZJLORAAXDAJLDC-CQDKDKBSSA-N 0.000 description 1
- XKXAZPSREVUCRT-BPNCWPANSA-N Ala-Tyr-Met Chemical compound CSCC[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)[C@H](C)N)CC1=CC=C(O)C=C1 XKXAZPSREVUCRT-BPNCWPANSA-N 0.000 description 1
- REWSWYIDQIELBE-FXQIFTODSA-N Ala-Val-Ser Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CO)C(O)=O REWSWYIDQIELBE-FXQIFTODSA-N 0.000 description 1
- ZDILXFDENZVOTL-BPNCWPANSA-N Ala-Val-Tyr Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O ZDILXFDENZVOTL-BPNCWPANSA-N 0.000 description 1
- 244000303258 Annona diversifolia Species 0.000 description 1
- 235000002198 Annona diversifolia Nutrition 0.000 description 1
- SGYSTDWPNPKJPP-GUBZILKMSA-N Arg-Ala-Arg Chemical compound NC(=N)NCCC[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O SGYSTDWPNPKJPP-GUBZILKMSA-N 0.000 description 1
- UXJCMQFPDWCHKX-DCAQKATOSA-N Arg-Arg-Glu Chemical compound NC(N)=NCCC[C@H](N)C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CCC(O)=O)C(O)=O UXJCMQFPDWCHKX-DCAQKATOSA-N 0.000 description 1
- HJVGMOYJDDXLMI-AVGNSLFASA-N Arg-Arg-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@@H](N)CCCNC(N)=N HJVGMOYJDDXLMI-AVGNSLFASA-N 0.000 description 1
- KMSHNDWHPWXPEC-BQBZGAKWSA-N Arg-Asp-Gly Chemical compound NC(N)=NCCC[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)NCC(O)=O KMSHNDWHPWXPEC-BQBZGAKWSA-N 0.000 description 1
- OBFTYSPXDRROQO-SRVKXCTJSA-N Arg-Gln-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@H](CCC(N)=O)NC(=O)[C@@H](N)CCCN=C(N)N OBFTYSPXDRROQO-SRVKXCTJSA-N 0.000 description 1
- ZEAYJGRKRUBDOB-GARJFASQSA-N Arg-Gln-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CCCN=C(N)N)N)C(=O)O ZEAYJGRKRUBDOB-GARJFASQSA-N 0.000 description 1
- UFBURHXMKFQVLM-CIUDSAMLSA-N Arg-Glu-Ser Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CO)C(O)=O UFBURHXMKFQVLM-CIUDSAMLSA-N 0.000 description 1
- PHHRSPBBQUFULD-UWVGGRQHSA-N Arg-Gly-Lys Chemical compound C(CCN)C[C@@H](C(=O)O)NC(=O)CNC(=O)[C@H](CCCN=C(N)N)N PHHRSPBBQUFULD-UWVGGRQHSA-N 0.000 description 1
- FNXCAFKDGBROCU-STECZYCISA-N Arg-Ile-Tyr Chemical compound NC(N)=NCCC[C@H](N)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 FNXCAFKDGBROCU-STECZYCISA-N 0.000 description 1
- WMEVEPXNCMKNGH-IHRRRGAJSA-N Arg-Leu-His Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)NC(=O)[C@H](CCCN=C(N)N)N WMEVEPXNCMKNGH-IHRRRGAJSA-N 0.000 description 1
- COXMUHNBYCVVRG-DCAQKATOSA-N Arg-Leu-Ser Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(O)=O COXMUHNBYCVVRG-DCAQKATOSA-N 0.000 description 1
- JEOCWTUOMKEEMF-RHYQMDGZSA-N Arg-Leu-Thr Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O JEOCWTUOMKEEMF-RHYQMDGZSA-N 0.000 description 1
- AOHKLEBWKMKITA-IHRRRGAJSA-N Arg-Phe-Ser Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CO)C(=O)O)NC(=O)[C@H](CCCN=C(N)N)N AOHKLEBWKMKITA-IHRRRGAJSA-N 0.000 description 1
- BSYKSCBTTQKOJG-GUBZILKMSA-N Arg-Pro-Ala Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](C)C(O)=O BSYKSCBTTQKOJG-GUBZILKMSA-N 0.000 description 1
- DNBMCNQKNOKOSD-DCAQKATOSA-N Arg-Pro-Gln Chemical compound NC(N)=NCCC[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CCC(N)=O)C(O)=O DNBMCNQKNOKOSD-DCAQKATOSA-N 0.000 description 1
- LRPZJPMQGKGHSG-XGEHTFHBSA-N Arg-Ser-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CO)NC(=O)[C@H](CCCN=C(N)N)N)O LRPZJPMQGKGHSG-XGEHTFHBSA-N 0.000 description 1
- FBXMCPLCVYUWBO-BPUTZDHNSA-N Arg-Ser-Trp Chemical compound C1=CC=C2C(=C1)C(=CN2)C[C@@H](C(=O)O)NC(=O)[C@H](CO)NC(=O)[C@H](CCCN=C(N)N)N FBXMCPLCVYUWBO-BPUTZDHNSA-N 0.000 description 1
- OQPAZKMGCWPERI-GUBZILKMSA-N Arg-Ser-Val Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(O)=O OQPAZKMGCWPERI-GUBZILKMSA-N 0.000 description 1
- QJWLLRZTJFPCHA-STECZYCISA-N Arg-Tyr-Ile Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O QJWLLRZTJFPCHA-STECZYCISA-N 0.000 description 1
- HUZGPXBILPMCHM-IHRRRGAJSA-N Asn-Arg-Phe Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O HUZGPXBILPMCHM-IHRRRGAJSA-N 0.000 description 1
- QNJIRRVTOXNGMH-GUBZILKMSA-N Asn-Gln-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@H](CCC(N)=O)NC(=O)[C@@H](N)CC(N)=O QNJIRRVTOXNGMH-GUBZILKMSA-N 0.000 description 1
- JREOBWLIZLXRIS-GUBZILKMSA-N Asn-Glu-Leu Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(O)=O JREOBWLIZLXRIS-GUBZILKMSA-N 0.000 description 1
- DXVMJJNAOVECBA-WHFBIAKZSA-N Asn-Gly-Asn Chemical compound NC(=O)C[C@H](N)C(=O)NCC(=O)N[C@@H](CC(N)=O)C(O)=O DXVMJJNAOVECBA-WHFBIAKZSA-N 0.000 description 1
- HDHZCEDPLTVHFZ-GUBZILKMSA-N Asn-Leu-Glu Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(O)=O)C(O)=O HDHZCEDPLTVHFZ-GUBZILKMSA-N 0.000 description 1
- WIDVAWAQBRAKTI-YUMQZZPRSA-N Asn-Leu-Gly Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC(C)C)C(=O)NCC(O)=O WIDVAWAQBRAKTI-YUMQZZPRSA-N 0.000 description 1
- RCFGLXMZDYNRSC-CIUDSAMLSA-N Asn-Lys-Ala Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](C)C(O)=O RCFGLXMZDYNRSC-CIUDSAMLSA-N 0.000 description 1
- XMHFCUKJRCQXGI-CIUDSAMLSA-N Asn-Pro-Gln Chemical compound C1C[C@H](N(C1)C(=O)[C@H](CC(=O)N)N)C(=O)N[C@@H](CCC(=O)N)C(=O)O XMHFCUKJRCQXGI-CIUDSAMLSA-N 0.000 description 1
- XTMZYFMTYJNABC-ZLUOBGJFSA-N Asn-Ser-Ala Chemical compound C[C@@H](C(=O)O)NC(=O)[C@H](CO)NC(=O)[C@H](CC(=O)N)N XTMZYFMTYJNABC-ZLUOBGJFSA-N 0.000 description 1
- MKJBPDLENBUHQU-CIUDSAMLSA-N Asn-Ser-Leu Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(O)=O MKJBPDLENBUHQU-CIUDSAMLSA-N 0.000 description 1
- JBDLMLZNDRLDIX-HJGDQZAQSA-N Asn-Thr-Leu Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(C)C)C(O)=O JBDLMLZNDRLDIX-HJGDQZAQSA-N 0.000 description 1
- AMGQTNHANMRPOE-LKXGYXEUSA-N Asn-Thr-Ser Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CO)C(O)=O AMGQTNHANMRPOE-LKXGYXEUSA-N 0.000 description 1
- LGCVSPFCFXWUEY-IHPCNDPISA-N Asn-Trp-Tyr Chemical compound C1=CC=C2C(=C1)C(=CN2)C[C@@H](C(=O)N[C@@H](CC3=CC=C(C=C3)O)C(=O)O)NC(=O)[C@H](CC(=O)N)N LGCVSPFCFXWUEY-IHPCNDPISA-N 0.000 description 1
- JPPLRQVZMZFOSX-UWJYBYFXSA-N Asn-Tyr-Ala Chemical compound NC(=O)C[C@H](N)C(=O)N[C@H](C(=O)N[C@@H](C)C(O)=O)CC1=CC=C(O)C=C1 JPPLRQVZMZFOSX-UWJYBYFXSA-N 0.000 description 1
- DATSKXOXPUAOLK-KKUMJFAQSA-N Asn-Tyr-Leu Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(C)C)C(O)=O DATSKXOXPUAOLK-KKUMJFAQSA-N 0.000 description 1
- DPWDPEVGACCWTC-SRVKXCTJSA-N Asn-Tyr-Ser Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CO)C(O)=O DPWDPEVGACCWTC-SRVKXCTJSA-N 0.000 description 1
- CBWCQCANJSGUOH-ZKWXMUAHSA-N Asn-Val-Ala Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](C)C(O)=O CBWCQCANJSGUOH-ZKWXMUAHSA-N 0.000 description 1
- XPGVTUBABLRGHY-BIIVOSGPSA-N Asp-Ala-Pro Chemical compound C[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CC(=O)O)N XPGVTUBABLRGHY-BIIVOSGPSA-N 0.000 description 1
- CNKAZIGBGQIHLL-GUBZILKMSA-N Asp-Arg-Met Chemical compound CSCC[C@@H](C(=O)O)NC(=O)[C@H](CCCN=C(N)N)NC(=O)[C@H](CC(=O)O)N CNKAZIGBGQIHLL-GUBZILKMSA-N 0.000 description 1
- MRQQMVZUHXUPEV-IHRRRGAJSA-N Asp-Arg-Phe Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O MRQQMVZUHXUPEV-IHRRRGAJSA-N 0.000 description 1
- XYBJLTKSGFBLCS-QXEWZRGKSA-N Asp-Arg-Val Chemical compound NC(N)=NCCC[C@@H](C(=O)N[C@@H](C(C)C)C(O)=O)NC(=O)[C@@H](N)CC(O)=O XYBJLTKSGFBLCS-QXEWZRGKSA-N 0.000 description 1
- KNMRXHIAVXHCLW-ZLUOBGJFSA-N Asp-Asn-Ser Chemical compound C([C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H](CO)C(=O)O)N)C(=O)O KNMRXHIAVXHCLW-ZLUOBGJFSA-N 0.000 description 1
- BFOYULZBKYOKAN-OLHMAJIHSA-N Asp-Asp-Thr Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O BFOYULZBKYOKAN-OLHMAJIHSA-N 0.000 description 1
- KHBLRHKVXICFMY-GUBZILKMSA-N Asp-Glu-Lys Chemical compound N[C@@H](CC(=O)O)C(=O)N[C@@H](CCC(=O)O)C(=O)N[C@@H](CCCCN)C(=O)O KHBLRHKVXICFMY-GUBZILKMSA-N 0.000 description 1
- QCVXMEHGFUMKCO-YUMQZZPRSA-N Asp-Gly-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)CNC(=O)[C@@H](N)CC(O)=O QCVXMEHGFUMKCO-YUMQZZPRSA-N 0.000 description 1
- KTTCQQNRRLCIBC-GHCJXIJMSA-N Asp-Ile-Ala Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](C)C(O)=O KTTCQQNRRLCIBC-GHCJXIJMSA-N 0.000 description 1
- QNFRBNZGVVKBNJ-PEFMBERDSA-N Asp-Ile-Gln Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)O)NC(=O)[C@H](CC(=O)O)N QNFRBNZGVVKBNJ-PEFMBERDSA-N 0.000 description 1
- KLYPOCBLKMPBIQ-GHCJXIJMSA-N Asp-Ile-Ser Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CO)C(=O)O)NC(=O)[C@H](CC(=O)O)N KLYPOCBLKMPBIQ-GHCJXIJMSA-N 0.000 description 1
- UZNSWMFLKVKJLI-VHWLVUOQSA-N Asp-Ile-Trp Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(O)=O UZNSWMFLKVKJLI-VHWLVUOQSA-N 0.000 description 1
- FQHBAQLBIXLWAG-DCAQKATOSA-N Asp-Lys-Met Chemical compound CSCC[C@@H](C(=O)O)NC(=O)[C@H](CCCCN)NC(=O)[C@H](CC(=O)O)N FQHBAQLBIXLWAG-DCAQKATOSA-N 0.000 description 1
- DPNWSMBUYCLEDG-CIUDSAMLSA-N Asp-Lys-Ser Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CO)C(O)=O DPNWSMBUYCLEDG-CIUDSAMLSA-N 0.000 description 1
- USNJAPJZSGTTPX-XVSYOHENSA-N Asp-Phe-Thr Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H]([C@@H](C)O)C(O)=O USNJAPJZSGTTPX-XVSYOHENSA-N 0.000 description 1
- MNQMTYSEKZHIDF-GCJQMDKQSA-N Asp-Thr-Ala Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C)C(O)=O MNQMTYSEKZHIDF-GCJQMDKQSA-N 0.000 description 1
- ITGFVUYOLWBPQW-KKHAAJSZSA-N Asp-Thr-Val Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C(C)C)C(O)=O ITGFVUYOLWBPQW-KKHAAJSZSA-N 0.000 description 1
- GHAHOJDCBRXAKC-IHPCNDPISA-N Asp-Trp-Tyr Chemical compound C1=CC=C2C(=C1)C(=CN2)C[C@@H](C(=O)N[C@@H](CC3=CC=C(C=C3)O)C(=O)O)NC(=O)[C@H](CC(=O)O)N GHAHOJDCBRXAKC-IHPCNDPISA-N 0.000 description 1
- CZIVKMOEXPILDK-SRVKXCTJSA-N Asp-Tyr-Ser Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CO)C(O)=O CZIVKMOEXPILDK-SRVKXCTJSA-N 0.000 description 1
- XWKBWZXGNXTDKY-ZKWXMUAHSA-N Asp-Val-Ala Chemical compound OC(=O)[C@H](C)NC(=O)[C@H](C(C)C)NC(=O)[C@@H](N)CC(O)=O XWKBWZXGNXTDKY-ZKWXMUAHSA-N 0.000 description 1
- 241000714230 Avian leukemia virus Species 0.000 description 1
- 230000003844 B-cell-activation Effects 0.000 description 1
- 229940124290 BCR-ABL tyrosine kinase inhibitor Drugs 0.000 description 1
- 241000145903 Bombyx mori cypovirus 1 Species 0.000 description 1
- 208000003174 Brain Neoplasms Diseases 0.000 description 1
- 208000025821 CD4 deficiency Diseases 0.000 description 1
- 241000282465 Canis Species 0.000 description 1
- 208000005623 Carcinogenesis Diseases 0.000 description 1
- 201000009030 Carcinoma Diseases 0.000 description 1
- 241001466804 Carnivora Species 0.000 description 1
- 102000000844 Cell Surface Receptors Human genes 0.000 description 1
- 108010001857 Cell Surface Receptors Proteins 0.000 description 1
- 206010008583 Chloroma Diseases 0.000 description 1
- 108091062157 Cis-regulatory element Proteins 0.000 description 1
- 206010010356 Congenital anomaly Diseases 0.000 description 1
- 102000004420 Creatine Kinase Human genes 0.000 description 1
- 108010042126 Creatine kinase Proteins 0.000 description 1
- 241000699800 Cricetinae Species 0.000 description 1
- TVYMKYUSZSVOAG-ZLUOBGJFSA-N Cys-Ala-Ala Chemical compound [H]N[C@@H](CS)C(=O)N[C@@H](C)C(=O)N[C@@H](C)C(O)=O TVYMKYUSZSVOAG-ZLUOBGJFSA-N 0.000 description 1
- AMRLSQGGERHDHJ-FXQIFTODSA-N Cys-Ala-Arg Chemical compound [H]N[C@@H](CS)C(=O)N[C@@H](C)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O AMRLSQGGERHDHJ-FXQIFTODSA-N 0.000 description 1
- QLCPDGRAEJSYQM-LPEHRKFASA-N Cys-Arg-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CCCN=C(N)N)NC(=O)[C@H](CS)N)C(=O)O QLCPDGRAEJSYQM-LPEHRKFASA-N 0.000 description 1
- GEEXORWTBTUOHC-FXQIFTODSA-N Cys-Arg-Ser Chemical compound C(C[C@@H](C(=O)N[C@@H](CO)C(=O)O)NC(=O)[C@H](CS)N)CN=C(N)N GEEXORWTBTUOHC-FXQIFTODSA-N 0.000 description 1
- CPTUXCUWQIBZIF-ZLUOBGJFSA-N Cys-Asn-Ser Chemical compound SC[C@H](N)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CO)C(O)=O CPTUXCUWQIBZIF-ZLUOBGJFSA-N 0.000 description 1
- WDQXKVCQXRNOSI-GHCJXIJMSA-N Cys-Asp-Ile Chemical compound [H]N[C@@H](CS)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O WDQXKVCQXRNOSI-GHCJXIJMSA-N 0.000 description 1
- BPHKULHWEIUDOB-FXQIFTODSA-N Cys-Gln-Gln Chemical compound SC[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCC(N)=O)C(O)=O BPHKULHWEIUDOB-FXQIFTODSA-N 0.000 description 1
- UXIYYUMGFNSGBK-XPUUQOCRSA-N Cys-Gly-Val Chemical compound [H]N[C@@H](CS)C(=O)NCC(=O)N[C@@H](C(C)C)C(O)=O UXIYYUMGFNSGBK-XPUUQOCRSA-N 0.000 description 1
- ZXCAQANTQWBICD-DCAQKATOSA-N Cys-Lys-Val Chemical compound CC(C)[C@@H](C(=O)O)NC(=O)[C@H](CCCCN)NC(=O)[C@H](CS)N ZXCAQANTQWBICD-DCAQKATOSA-N 0.000 description 1
- CHRCKSPMGYDLIA-SRVKXCTJSA-N Cys-Phe-Ser Chemical compound [H]N[C@@H](CS)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CO)C(O)=O CHRCKSPMGYDLIA-SRVKXCTJSA-N 0.000 description 1
- KFYPRIGJTICABD-XGEHTFHBSA-N Cys-Thr-Val Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](C(C)C)C(=O)O)NC(=O)[C@H](CS)N)O KFYPRIGJTICABD-XGEHTFHBSA-N 0.000 description 1
- KZZYVYWSXMFYEC-DCAQKATOSA-N Cys-Val-Leu Chemical compound [H]N[C@@H](CS)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CC(C)C)C(O)=O KZZYVYWSXMFYEC-DCAQKATOSA-N 0.000 description 1
- 241000252212 Danio rerio Species 0.000 description 1
- BWGNESOTFCXPMA-UHFFFAOYSA-N Dihydrogen disulfide Chemical compound SS BWGNESOTFCXPMA-UHFFFAOYSA-N 0.000 description 1
- 206010061818 Disease progression Diseases 0.000 description 1
- 206010061819 Disease recurrence Diseases 0.000 description 1
- 102000001301 EGF receptor Human genes 0.000 description 1
- 108060006698 EGF receptor Proteins 0.000 description 1
- 101150039808 Egfr gene Proteins 0.000 description 1
- 108010074864 Factor XI Proteins 0.000 description 1
- 206010053717 Fibrous histiocytoma Diseases 0.000 description 1
- 241000710198 Foot-and-mouth disease virus Species 0.000 description 1
- 101000834253 Gallus gallus Actin, cytoplasmic 1 Proteins 0.000 description 1
- 208000021309 Germ cell tumor Diseases 0.000 description 1
- 208000007569 Giant Cell Tumors Diseases 0.000 description 1
- RZSLYUUFFVHFRQ-FXQIFTODSA-N Gln-Ala-Glu Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](C)C(=O)N[C@@H](CCC(O)=O)C(O)=O RZSLYUUFFVHFRQ-FXQIFTODSA-N 0.000 description 1
- KVYVOGYEMPEXBT-GUBZILKMSA-N Gln-Ala-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](C)NC(=O)[C@@H](N)CCC(N)=O KVYVOGYEMPEXBT-GUBZILKMSA-N 0.000 description 1
- KWUSGAIFNHQCBY-DCAQKATOSA-N Gln-Arg-Arg Chemical compound NC(=O)CC[C@H](N)C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CCCN=C(N)N)C(O)=O KWUSGAIFNHQCBY-DCAQKATOSA-N 0.000 description 1
- QFJPFPCSXOXMKI-BPUTZDHNSA-N Gln-Gln-Trp Chemical compound C1=CC=C2C(=C1)C(=CN2)C[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CCC(=O)N)N QFJPFPCSXOXMKI-BPUTZDHNSA-N 0.000 description 1
- CGVWDTRDPLOMHZ-FXQIFTODSA-N Gln-Glu-Asp Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(O)=O)C(O)=O CGVWDTRDPLOMHZ-FXQIFTODSA-N 0.000 description 1
- KCJJFESQRXGTGC-BQBZGAKWSA-N Gln-Glu-Gly Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCC(O)=O)C(=O)NCC(O)=O KCJJFESQRXGTGC-BQBZGAKWSA-N 0.000 description 1
- ZNZPKVQURDQFFS-FXQIFTODSA-N Gln-Glu-Ser Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CO)C(O)=O ZNZPKVQURDQFFS-FXQIFTODSA-N 0.000 description 1
- XKBASPWPBXNVLQ-WDSKDSINSA-N Gln-Gly-Asn Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)NCC(=O)N[C@@H](CC(N)=O)C(O)=O XKBASPWPBXNVLQ-WDSKDSINSA-N 0.000 description 1
- FGYPOQPQTUNESW-IUCAKERBSA-N Gln-Gly-Leu Chemical compound CC(C)C[C@@H](C(=O)O)NC(=O)CNC(=O)[C@H](CCC(=O)N)N FGYPOQPQTUNESW-IUCAKERBSA-N 0.000 description 1
- JXFLPKSDLDEOQK-JHEQGTHGSA-N Gln-Gly-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)CNC(=O)[C@@H](N)CCC(N)=O JXFLPKSDLDEOQK-JHEQGTHGSA-N 0.000 description 1
- LGIKBBLQVSWUGK-DCAQKATOSA-N Gln-Leu-Gln Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(N)=O)C(O)=O LGIKBBLQVSWUGK-DCAQKATOSA-N 0.000 description 1
- YPMDZWPZFOZYFG-GUBZILKMSA-N Gln-Leu-Ser Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(O)=O YPMDZWPZFOZYFG-GUBZILKMSA-N 0.000 description 1
- TWIAMTNJOMRDAK-GUBZILKMSA-N Gln-Lys-Asp Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(O)=O)C(O)=O TWIAMTNJOMRDAK-GUBZILKMSA-N 0.000 description 1
- MSHXWFKYXJTLEZ-CIUDSAMLSA-N Gln-Met-Asn Chemical compound CSCC[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)O)NC(=O)[C@H](CCC(=O)N)N MSHXWFKYXJTLEZ-CIUDSAMLSA-N 0.000 description 1
- ROHVCXBMIAAASL-HJGDQZAQSA-N Gln-Met-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CCSC)NC(=O)[C@H](CCC(=O)N)N)O ROHVCXBMIAAASL-HJGDQZAQSA-N 0.000 description 1
- HMIXCETWRYDVMO-GUBZILKMSA-N Gln-Pro-Glu Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CCC(O)=O)C(O)=O HMIXCETWRYDVMO-GUBZILKMSA-N 0.000 description 1
- FQCILXROGNOZON-YUMQZZPRSA-N Gln-Pro-Gly Chemical compound NC(=O)CC[C@H](N)C(=O)N1CCC[C@H]1C(=O)NCC(O)=O FQCILXROGNOZON-YUMQZZPRSA-N 0.000 description 1
- XQDGOJPVMSWZSO-SRVKXCTJSA-N Gln-Pro-Leu Chemical compound CC(C)C[C@@H](C(=O)O)NC(=O)[C@@H]1CCCN1C(=O)[C@H](CCC(=O)N)N XQDGOJPVMSWZSO-SRVKXCTJSA-N 0.000 description 1
- OSCLNNWLKKIQJM-WDSKDSINSA-N Gln-Ser-Gly Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CO)C(=O)NCC(O)=O OSCLNNWLKKIQJM-WDSKDSINSA-N 0.000 description 1
- LPIKVBWNNVFHCQ-GUBZILKMSA-N Gln-Ser-Leu Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(O)=O LPIKVBWNNVFHCQ-GUBZILKMSA-N 0.000 description 1
- VOUSELYGTNGEPB-NUMRIWBASA-N Gln-Thr-Asp Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(O)=O)C(O)=O VOUSELYGTNGEPB-NUMRIWBASA-N 0.000 description 1
- STHSGOZLFLFGSS-SUSMZKCASA-N Gln-Thr-Thr Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O STHSGOZLFLFGSS-SUSMZKCASA-N 0.000 description 1
- HPBKQFJXDUVNQV-FHWLQOOXSA-N Gln-Tyr-Tyr Chemical compound C1=CC(=CC=C1C[C@@H](C(=O)N[C@@H](CC2=CC=C(C=C2)O)C(=O)O)NC(=O)[C@H](CCC(=O)N)N)O HPBKQFJXDUVNQV-FHWLQOOXSA-N 0.000 description 1
- KBKGRMNVKPSQIF-XDTLVQLUSA-N Glu-Ala-Tyr Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O KBKGRMNVKPSQIF-XDTLVQLUSA-N 0.000 description 1
- WOMUDRVDJMHTCV-DCAQKATOSA-N Glu-Arg-Arg Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O WOMUDRVDJMHTCV-DCAQKATOSA-N 0.000 description 1
- ZOXBSICWUDAOHX-GUBZILKMSA-N Glu-Asn-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@@H](N)CCC(O)=O ZOXBSICWUDAOHX-GUBZILKMSA-N 0.000 description 1
- JVSBYEDSSRZQGV-GUBZILKMSA-N Glu-Asp-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CC(O)=O)NC(=O)[C@@H](N)CCC(O)=O JVSBYEDSSRZQGV-GUBZILKMSA-N 0.000 description 1
- JRCUFCXYZLPSDZ-ACZMJKKPSA-N Glu-Asp-Ser Chemical compound OC(=O)CC[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CO)C(O)=O JRCUFCXYZLPSDZ-ACZMJKKPSA-N 0.000 description 1
- SBCYJMOOHUDWDA-NUMRIWBASA-N Glu-Asp-Thr Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O SBCYJMOOHUDWDA-NUMRIWBASA-N 0.000 description 1
- XHUCVVHRLNPZSZ-CIUDSAMLSA-N Glu-Gln-Glu Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCC(O)=O)C(O)=O XHUCVVHRLNPZSZ-CIUDSAMLSA-N 0.000 description 1
- SJPMNHCEWPTRBR-BQBZGAKWSA-N Glu-Glu-Gly Chemical compound OC(=O)CC[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(=O)NCC(O)=O SJPMNHCEWPTRBR-BQBZGAKWSA-N 0.000 description 1
- MUSGDMDGNGXULI-DCAQKATOSA-N Glu-Glu-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@@H](N)CCC(O)=O MUSGDMDGNGXULI-DCAQKATOSA-N 0.000 description 1
- VXQOONWNIWFOCS-HGNGGELXSA-N Glu-His-Ala Chemical compound C[C@@H](C(=O)O)NC(=O)[C@H](CC1=CN=CN1)NC(=O)[C@H](CCC(=O)O)N VXQOONWNIWFOCS-HGNGGELXSA-N 0.000 description 1
- ITBHUUMCJJQUSC-LAEOZQHASA-N Glu-Ile-Gly Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H]([C@@H](C)CC)C(=O)NCC(O)=O ITBHUUMCJJQUSC-LAEOZQHASA-N 0.000 description 1
- XTZDZAXYPDISRR-MNXVOIDGSA-N Glu-Ile-Lys Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CCC(=O)O)N XTZDZAXYPDISRR-MNXVOIDGSA-N 0.000 description 1
- SWRVAQHFBRZVNX-GUBZILKMSA-N Glu-Lys-Asn Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(N)=O)C(O)=O SWRVAQHFBRZVNX-GUBZILKMSA-N 0.000 description 1
- CUPSDFQZTVVTSK-GUBZILKMSA-N Glu-Lys-Asp Chemical compound OC(=O)C[C@@H](C(O)=O)NC(=O)[C@H](CCCCN)NC(=O)[C@@H](N)CCC(O)=O CUPSDFQZTVVTSK-GUBZILKMSA-N 0.000 description 1
- FGSGPLRPQCZBSQ-AVGNSLFASA-N Glu-Phe-Ser Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CO)C(O)=O FGSGPLRPQCZBSQ-AVGNSLFASA-N 0.000 description 1
- RFTVTKBHDXCEEX-WDSKDSINSA-N Glu-Ser-Gly Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CO)C(=O)NCC(O)=O RFTVTKBHDXCEEX-WDSKDSINSA-N 0.000 description 1
- HGJREIGJLUQBTJ-SZMVWBNQSA-N Glu-Trp-Leu Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(=O)N[C@@H](CC(C)C)C(O)=O HGJREIGJLUQBTJ-SZMVWBNQSA-N 0.000 description 1
- CGWHAXBNGYQBBK-JBACZVJFSA-N Glu-Trp-Tyr Chemical compound C([C@H](NC(=O)[C@H](CC=1C2=CC=CC=C2NC=1)NC(=O)[C@H](CCC(O)=O)N)C(O)=O)C1=CC=C(O)C=C1 CGWHAXBNGYQBBK-JBACZVJFSA-N 0.000 description 1
- FGGKGJHCVMYGCD-UKJIMTQDSA-N Glu-Val-Ile Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O FGGKGJHCVMYGCD-UKJIMTQDSA-N 0.000 description 1
- LJPIRKICOISLKN-WHFBIAKZSA-N Gly-Ala-Ser Chemical compound NCC(=O)N[C@@H](C)C(=O)N[C@@H](CO)C(O)=O LJPIRKICOISLKN-WHFBIAKZSA-N 0.000 description 1
- JRDYDYXZKFNNRQ-XPUUQOCRSA-N Gly-Ala-Val Chemical compound CC(C)[C@@H](C(O)=O)NC(=O)[C@H](C)NC(=O)CN JRDYDYXZKFNNRQ-XPUUQOCRSA-N 0.000 description 1
- KFMBRBPXHVMDFN-UWVGGRQHSA-N Gly-Arg-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)CN)CCCNC(N)=N KFMBRBPXHVMDFN-UWVGGRQHSA-N 0.000 description 1
- KRRMJKMGWWXWDW-STQMWFEESA-N Gly-Arg-Phe Chemical compound NC(=N)NCCC[C@H](NC(=O)CN)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 KRRMJKMGWWXWDW-STQMWFEESA-N 0.000 description 1
- GGEJHJIXRBTJPD-BYPYZUCNSA-N Gly-Asn-Gly Chemical compound NCC(=O)N[C@@H](CC(N)=O)C(=O)NCC(O)=O GGEJHJIXRBTJPD-BYPYZUCNSA-N 0.000 description 1
- IWAXHBCACVWNHT-BQBZGAKWSA-N Gly-Asp-Arg Chemical compound NCC(=O)N[C@@H](CC(O)=O)C(=O)N[C@H](C(O)=O)CCCN=C(N)N IWAXHBCACVWNHT-BQBZGAKWSA-N 0.000 description 1
- PMNHJLASAAWELO-FOHZUACHSA-N Gly-Asp-Thr Chemical compound [H]NCC(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O PMNHJLASAAWELO-FOHZUACHSA-N 0.000 description 1
- YZACQYVWLCQWBT-BQBZGAKWSA-N Gly-Cys-Arg Chemical compound [H]NCC(=O)N[C@@H](CS)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O YZACQYVWLCQWBT-BQBZGAKWSA-N 0.000 description 1
- XXGQRGQPGFYECI-WDSKDSINSA-N Gly-Cys-Glu Chemical compound NCC(=O)N[C@@H](CS)C(=O)N[C@H](C(O)=O)CCC(O)=O XXGQRGQPGFYECI-WDSKDSINSA-N 0.000 description 1
- BYYNJRSNDARRBX-YFKPBYRVSA-N Gly-Gln-Gly Chemical compound NCC(=O)N[C@@H](CCC(N)=O)C(=O)NCC(O)=O BYYNJRSNDARRBX-YFKPBYRVSA-N 0.000 description 1
- GNPVTZJUUBPZKW-WDSKDSINSA-N Gly-Gln-Ser Chemical compound [H]NCC(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CO)C(O)=O GNPVTZJUUBPZKW-WDSKDSINSA-N 0.000 description 1
- DHDOADIPGZTAHT-YUMQZZPRSA-N Gly-Glu-Arg Chemical compound NCC(=O)N[C@@H](CCC(O)=O)C(=O)N[C@H](C(O)=O)CCCN=C(N)N DHDOADIPGZTAHT-YUMQZZPRSA-N 0.000 description 1
- UQJNXZSSGQIPIQ-FBCQKBJTSA-N Gly-Gly-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)CNC(=O)CN UQJNXZSSGQIPIQ-FBCQKBJTSA-N 0.000 description 1
- VAXIVIPMCTYSHI-YUMQZZPRSA-N Gly-His-Asp Chemical compound C1=C(NC=N1)C[C@@H](C(=O)N[C@@H](CC(=O)O)C(=O)O)NC(=O)CN VAXIVIPMCTYSHI-YUMQZZPRSA-N 0.000 description 1
- ITZOBNKQDZEOCE-NHCYSSNCSA-N Gly-Ile-Lys Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)CN ITZOBNKQDZEOCE-NHCYSSNCSA-N 0.000 description 1
- NNCSJUBVFBDDLC-YUMQZZPRSA-N Gly-Leu-Ser Chemical compound NCC(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(O)=O NNCSJUBVFBDDLC-YUMQZZPRSA-N 0.000 description 1
- PDUHNKAFQXQNLH-ZETCQYMHSA-N Gly-Lys-Gly Chemical compound NCCCC[C@H](NC(=O)CN)C(=O)NCC(O)=O PDUHNKAFQXQNLH-ZETCQYMHSA-N 0.000 description 1
- YLEIWGJJBFBFHC-KBPBESRZSA-N Gly-Phe-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)CN)CC1=CC=CC=C1 YLEIWGJJBFBFHC-KBPBESRZSA-N 0.000 description 1
- NSVOVKWEKGEOQB-LURJTMIESA-N Gly-Pro-Gly Chemical compound NCC(=O)N1CCC[C@H]1C(=O)NCC(O)=O NSVOVKWEKGEOQB-LURJTMIESA-N 0.000 description 1
- CSMYMGFCEJWALV-WDSKDSINSA-N Gly-Ser-Gln Chemical compound NCC(=O)N[C@@H](CO)C(=O)N[C@H](C(O)=O)CCC(N)=O CSMYMGFCEJWALV-WDSKDSINSA-N 0.000 description 1
- WCORRBXVISTKQL-WHFBIAKZSA-N Gly-Ser-Ser Chemical compound NCC(=O)N[C@@H](CO)C(=O)N[C@@H](CO)C(O)=O WCORRBXVISTKQL-WHFBIAKZSA-N 0.000 description 1
- NVTPVQLIZCOJFK-FOHZUACHSA-N Gly-Thr-Asp Chemical compound [H]NCC(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(O)=O)C(O)=O NVTPVQLIZCOJFK-FOHZUACHSA-N 0.000 description 1
- ZZWUYQXMIFTIIY-WEDXCCLWSA-N Gly-Thr-Leu Chemical compound [H]NCC(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(C)C)C(O)=O ZZWUYQXMIFTIIY-WEDXCCLWSA-N 0.000 description 1
- TVTZEOHWHUVYCG-KYNKHSRBSA-N Gly-Thr-Thr Chemical compound [H]NCC(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O TVTZEOHWHUVYCG-KYNKHSRBSA-N 0.000 description 1
- CUVBTVWFVIIDOC-YEPSODPASA-N Gly-Thr-Val Chemical compound CC(C)[C@@H](C(O)=O)NC(=O)[C@H]([C@@H](C)O)NC(=O)CN CUVBTVWFVIIDOC-YEPSODPASA-N 0.000 description 1
- KSOBNUBCYHGUKH-UWVGGRQHSA-N Gly-Val-Val Chemical compound CC(C)[C@@H](C(O)=O)NC(=O)[C@H](C(C)C)NC(=O)CN KSOBNUBCYHGUKH-UWVGGRQHSA-N 0.000 description 1
- 208000024869 Goodpasture syndrome Diseases 0.000 description 1
- 102000009465 Growth Factor Receptors Human genes 0.000 description 1
- 108010009202 Growth Factor Receptors Proteins 0.000 description 1
- 239000007995 HEPES buffer Substances 0.000 description 1
- 102000001554 Hemoglobins Human genes 0.000 description 1
- 108010054147 Hemoglobins Proteins 0.000 description 1
- LSQHWKPPOFDHHZ-YUMQZZPRSA-N His-Asp-Gly Chemical compound C1=C(NC=N1)C[C@@H](C(=O)N[C@@H](CC(=O)O)C(=O)NCC(=O)O)N LSQHWKPPOFDHHZ-YUMQZZPRSA-N 0.000 description 1
- IWXMHXYOACDSIA-PYJNHQTQSA-N His-Ile-Val Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](C(C)C)C(O)=O IWXMHXYOACDSIA-PYJNHQTQSA-N 0.000 description 1
- NKRWVZQTPXPNRZ-SRVKXCTJSA-N His-Met-Gln Chemical compound NC(=O)CC[C@@H](C(O)=O)NC(=O)[C@H](CCSC)NC(=O)[C@@H](N)CC1=CN=CN1 NKRWVZQTPXPNRZ-SRVKXCTJSA-N 0.000 description 1
- YEKYGQZUBCRNGH-DCAQKATOSA-N His-Pro-Ser Chemical compound C1C[C@H](N(C1)C(=O)[C@H](CC2=CN=CN2)N)C(=O)N[C@@H](CO)C(=O)O YEKYGQZUBCRNGH-DCAQKATOSA-N 0.000 description 1
- ZHHLTWUOWXHVQJ-YUMQZZPRSA-N His-Ser-Gly Chemical compound C1=C(NC=N1)C[C@@H](C(=O)N[C@@H](CO)C(=O)NCC(=O)O)N ZHHLTWUOWXHVQJ-YUMQZZPRSA-N 0.000 description 1
- FCPSGEVYIVXPPO-QTKMDUPCSA-N His-Thr-Arg Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O FCPSGEVYIVXPPO-QTKMDUPCSA-N 0.000 description 1
- UWSMZKRTOZEGDD-CUJWVEQBSA-N His-Thr-Ser Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CO)C(O)=O UWSMZKRTOZEGDD-CUJWVEQBSA-N 0.000 description 1
- YERBCFWVWITTEJ-NAZCDGGXSA-N His-Trp-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CC1=CNC2=CC=CC=C21)NC(=O)[C@H](CC3=CN=CN3)N)O YERBCFWVWITTEJ-NAZCDGGXSA-N 0.000 description 1
- ZNTSGDNUITWTRA-WDSOQIARSA-N His-Trp-Val Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(=O)N[C@@H](C(C)C)C(O)=O ZNTSGDNUITWTRA-WDSOQIARSA-N 0.000 description 1
- HIJIJPFILYPTFR-ACRUOGEOSA-N His-Tyr-Tyr Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O HIJIJPFILYPTFR-ACRUOGEOSA-N 0.000 description 1
- GGXUJBKENKVYNV-ULQDDVLXSA-N His-Val-Phe Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)O)NC(=O)[C@H](CC2=CN=CN2)N GGXUJBKENKVYNV-ULQDDVLXSA-N 0.000 description 1
- 208000002291 Histiocytic Sarcoma Diseases 0.000 description 1
- 241001272567 Hominoidea Species 0.000 description 1
- 241000282412 Homo Species 0.000 description 1
- 101001002657 Homo sapiens Interleukin-2 Proteins 0.000 description 1
- 101100042692 Homo sapiens SLAMF7 gene Proteins 0.000 description 1
- 101000914484 Homo sapiens T-lymphocyte activation antigen CD80 Proteins 0.000 description 1
- 241000598436 Human T-cell lymphotropic virus Species 0.000 description 1
- VAXBXNPRXPHGHG-BJDJZHNGSA-N Ile-Ala-Leu Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](C)C(=O)N[C@@H](CC(C)C)C(=O)O)N VAXBXNPRXPHGHG-BJDJZHNGSA-N 0.000 description 1
- CYHYBSGMHMHKOA-CIQUZCHMSA-N Ile-Ala-Thr Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](C)C(=O)N[C@@H]([C@@H](C)O)C(=O)O)N CYHYBSGMHMHKOA-CIQUZCHMSA-N 0.000 description 1
- ASCFJMSGKUIRDU-ZPFDUUQYSA-N Ile-Arg-Gln Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(N)=O)C(O)=O ASCFJMSGKUIRDU-ZPFDUUQYSA-N 0.000 description 1
- UKTUOMWSJPXODT-GUDRVLHUSA-N Ile-Asn-Pro Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N1CCC[C@@H]1C(=O)O)N UKTUOMWSJPXODT-GUDRVLHUSA-N 0.000 description 1
- WNQKUUQIVDDAFA-ZPFDUUQYSA-N Ile-Gln-Met Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)N[C@@H](CCSC)C(=O)O)N WNQKUUQIVDDAFA-ZPFDUUQYSA-N 0.000 description 1
- YBJWJQQBWRARLT-KBIXCLLPSA-N Ile-Gln-Ser Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CO)C(O)=O YBJWJQQBWRARLT-KBIXCLLPSA-N 0.000 description 1
- UAQSZXGJGLHMNV-XEGUGMAKSA-N Ile-Gly-Tyr Chemical compound CC[C@H](C)[C@@H](C(=O)NCC(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)O)N UAQSZXGJGLHMNV-XEGUGMAKSA-N 0.000 description 1
- TWPSALMCEHCIOY-YTFOTSKYSA-N Ile-Ile-Leu Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC(C)C)C(=O)O)N TWPSALMCEHCIOY-YTFOTSKYSA-N 0.000 description 1
- CSQNHSGHAPRGPQ-YTFOTSKYSA-N Ile-Ile-Lys Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CCCCN)C(=O)O)N CSQNHSGHAPRGPQ-YTFOTSKYSA-N 0.000 description 1
- FZWVCYCYWCLQDH-NHCYSSNCSA-N Ile-Leu-Gly Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC(C)C)C(=O)NCC(=O)O)N FZWVCYCYWCLQDH-NHCYSSNCSA-N 0.000 description 1
- RMNMUUCYTMLWNA-ZPFDUUQYSA-N Ile-Lys-Asp Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(=O)O)C(=O)O)N RMNMUUCYTMLWNA-ZPFDUUQYSA-N 0.000 description 1
- ADDYYRVQQZFIMW-MNXVOIDGSA-N Ile-Lys-Glu Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCC(=O)O)C(=O)O)N ADDYYRVQQZFIMW-MNXVOIDGSA-N 0.000 description 1
- PARSHQDZROHERM-NHCYSSNCSA-N Ile-Lys-Gly Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)NCC(=O)O)N PARSHQDZROHERM-NHCYSSNCSA-N 0.000 description 1
- JZNVOBUNTWNZPW-GHCJXIJMSA-N Ile-Ser-Asp Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(=O)O)C(=O)O)N JZNVOBUNTWNZPW-GHCJXIJMSA-N 0.000 description 1
- FBGXMKUWQFPHFB-JBDRJPRFSA-N Ile-Ser-Cys Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CS)C(=O)O)N FBGXMKUWQFPHFB-JBDRJPRFSA-N 0.000 description 1
- VGSPNSSCMOHRRR-BJDJZHNGSA-N Ile-Ser-Lys Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CCCCN)C(=O)O)N VGSPNSSCMOHRRR-BJDJZHNGSA-N 0.000 description 1
- RKQAYOWLSFLJEE-SVSWQMSJSA-N Ile-Thr-Cys Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CS)C(=O)O)N RKQAYOWLSFLJEE-SVSWQMSJSA-N 0.000 description 1
- YBKKLDBBPFIXBQ-MBLNEYKQSA-N Ile-Thr-Gly Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H]([C@@H](C)O)C(=O)NCC(=O)O)N YBKKLDBBPFIXBQ-MBLNEYKQSA-N 0.000 description 1
- WKSHBPRUIRGWRZ-KCTSRDHCSA-N Ile-Trp-Gly Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC1=CNC2=CC=CC=C21)C(=O)NCC(=O)O)N WKSHBPRUIRGWRZ-KCTSRDHCSA-N 0.000 description 1
- DTPGSUQHUMELQB-GVARAGBVSA-N Ile-Tyr-Ala Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@H](C(=O)N[C@@H](C)C(O)=O)CC1=CC=C(O)C=C1 DTPGSUQHUMELQB-GVARAGBVSA-N 0.000 description 1
- REXAUQBGSGDEJY-IGISWZIWSA-N Ile-Tyr-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)N[C@@H]([C@@H](C)CC)C(=O)O)N REXAUQBGSGDEJY-IGISWZIWSA-N 0.000 description 1
- NXRNRBOKDBIVKQ-CXTHYWKRSA-N Ile-Tyr-Tyr Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)N[C@@H](CC2=CC=C(C=C2)O)C(=O)O)N NXRNRBOKDBIVKQ-CXTHYWKRSA-N 0.000 description 1
- UYODHPPSCXBNCS-XUXIUFHCSA-N Ile-Val-Leu Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@H](C(O)=O)CC(C)C UYODHPPSCXBNCS-XUXIUFHCSA-N 0.000 description 1
- ZSESFIFAYQEKRD-CYDGBPFRSA-N Ile-Val-Met Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCSC)C(=O)O)N ZSESFIFAYQEKRD-CYDGBPFRSA-N 0.000 description 1
- 102000006496 Immunoglobulin Heavy Chains Human genes 0.000 description 1
- 108010019476 Immunoglobulin Heavy Chains Proteins 0.000 description 1
- 102000010790 Interleukin-3 Receptors Human genes 0.000 description 1
- 108010038452 Interleukin-3 Receptors Proteins 0.000 description 1
- 102000010781 Interleukin-6 Receptors Human genes 0.000 description 1
- FADYJNXDPBKVCA-UHFFFAOYSA-N L-Phenylalanyl-L-lysin Natural products NCCCCC(C(O)=O)NC(=O)C(N)CC1=CC=CC=C1 FADYJNXDPBKVCA-UHFFFAOYSA-N 0.000 description 1
- UGTHTQWIQKEDEH-BQBZGAKWSA-N L-alanyl-L-prolylglycine zwitterion Chemical compound C[C@H](N)C(=O)N1CCC[C@H]1C(=O)NCC(O)=O UGTHTQWIQKEDEH-BQBZGAKWSA-N 0.000 description 1
- LHSGPCFBGJHPCY-UHFFFAOYSA-N L-leucine-L-tyrosine Natural products CC(C)CC(N)C(=O)NC(C(O)=O)CC1=CC=C(O)C=C1 LHSGPCFBGJHPCY-UHFFFAOYSA-N 0.000 description 1
- COLNVLDHVKWLRT-QMMMGPOBSA-N L-phenylalanine Chemical compound OC(=O)[C@@H](N)CC1=CC=CC=C1 COLNVLDHVKWLRT-QMMMGPOBSA-N 0.000 description 1
- QIVBCDIJIAJPQS-VIFPVBQESA-N L-tryptophane Chemical compound C1=CC=C2C(C[C@H](N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-VIFPVBQESA-N 0.000 description 1
- OUYCCCASQSFEME-QMMMGPOBSA-N L-tyrosine Chemical compound OC(=O)[C@@H](N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-QMMMGPOBSA-N 0.000 description 1
- LZDNBBYBDGBADK-UHFFFAOYSA-N L-valyl-L-tryptophan Natural products C1=CC=C2C(CC(NC(=O)C(N)C(C)C)C(O)=O)=CNC2=C1 LZDNBBYBDGBADK-UHFFFAOYSA-N 0.000 description 1
- WNGVUZWBXZKQES-YUMQZZPRSA-N Leu-Ala-Gly Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](C)C(=O)NCC(O)=O WNGVUZWBXZKQES-YUMQZZPRSA-N 0.000 description 1
- BQSLGJHIAGOZCD-CIUDSAMLSA-N Leu-Ala-Ser Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@@H](CO)C(O)=O BQSLGJHIAGOZCD-CIUDSAMLSA-N 0.000 description 1
- UCOCBWDBHCUPQP-DCAQKATOSA-N Leu-Arg-Ser Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CO)C(O)=O UCOCBWDBHCUPQP-DCAQKATOSA-N 0.000 description 1
- JKGHDYGZRDWHGA-SRVKXCTJSA-N Leu-Asn-Leu Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CC(C)C)C(O)=O JKGHDYGZRDWHGA-SRVKXCTJSA-N 0.000 description 1
- JQSXWJXBASFONF-KKUMJFAQSA-N Leu-Asp-Phe Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O JQSXWJXBASFONF-KKUMJFAQSA-N 0.000 description 1
- GBDMISNMNXVTNV-XIRDDKMYSA-N Leu-Asp-Trp Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(O)=O GBDMISNMNXVTNV-XIRDDKMYSA-N 0.000 description 1
- KAFOIVJDVSZUMD-UHFFFAOYSA-N Leu-Gln-Gln Natural products CC(C)CC(N)C(=O)NC(CCC(N)=O)C(=O)NC(CCC(N)=O)C(O)=O KAFOIVJDVSZUMD-UHFFFAOYSA-N 0.000 description 1
- FQZPTCNSNPWHLJ-AVGNSLFASA-N Leu-Gln-Lys Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCCCN)C(O)=O FQZPTCNSNPWHLJ-AVGNSLFASA-N 0.000 description 1
- CQGSYZCULZMEDE-UHFFFAOYSA-N Leu-Gln-Pro Natural products CC(C)CC(N)C(=O)NC(CCC(N)=O)C(=O)N1CCCC1C(O)=O CQGSYZCULZMEDE-UHFFFAOYSA-N 0.000 description 1
- CIVKXGPFXDIQBV-WDCWCFNPSA-N Leu-Gln-Thr Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O CIVKXGPFXDIQBV-WDCWCFNPSA-N 0.000 description 1
- HPBCTWSUJOGJSH-MNXVOIDGSA-N Leu-Glu-Ile Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O HPBCTWSUJOGJSH-MNXVOIDGSA-N 0.000 description 1
- QVFGXCVIXXBFHO-AVGNSLFASA-N Leu-Glu-Leu Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(O)=O QVFGXCVIXXBFHO-AVGNSLFASA-N 0.000 description 1
- LAPSXOAUPNOINL-YUMQZZPRSA-N Leu-Gly-Asp Chemical compound CC(C)C[C@H](N)C(=O)NCC(=O)N[C@H](C(O)=O)CC(O)=O LAPSXOAUPNOINL-YUMQZZPRSA-N 0.000 description 1
- KGCLIYGPQXUNLO-IUCAKERBSA-N Leu-Gly-Glu Chemical compound CC(C)C[C@H](N)C(=O)NCC(=O)N[C@H](C(O)=O)CCC(O)=O KGCLIYGPQXUNLO-IUCAKERBSA-N 0.000 description 1
- POZULHZYLPGXMR-ONGXEEELSA-N Leu-Gly-Val Chemical compound CC(C)C[C@H](N)C(=O)NCC(=O)N[C@@H](C(C)C)C(O)=O POZULHZYLPGXMR-ONGXEEELSA-N 0.000 description 1
- MPSBSKHOWJQHBS-IHRRRGAJSA-N Leu-His-Met Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)N[C@@H](CCSC)C(=O)O)N MPSBSKHOWJQHBS-IHRRRGAJSA-N 0.000 description 1
- DSFYPIUSAMSERP-IHRRRGAJSA-N Leu-Leu-Arg Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@H](C(O)=O)CCCN=C(N)N DSFYPIUSAMSERP-IHRRRGAJSA-N 0.000 description 1
- KYIIALJHAOIAHF-KKUMJFAQSA-N Leu-Leu-His Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@H](C(O)=O)CC1=CN=CN1 KYIIALJHAOIAHF-KKUMJFAQSA-N 0.000 description 1
- FAELBUXXFQLUAX-AJNGGQMLSA-N Leu-Leu-Ile Chemical compound CC[C@H](C)[C@@H](C(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@@H](N)CC(C)C FAELBUXXFQLUAX-AJNGGQMLSA-N 0.000 description 1
- LXKNSJLSGPNHSK-KKUMJFAQSA-N Leu-Leu-Lys Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(=O)O)N LXKNSJLSGPNHSK-KKUMJFAQSA-N 0.000 description 1
- DCGXHWINSHEPIR-SRVKXCTJSA-N Leu-Lys-Cys Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CS)C(=O)O)N DCGXHWINSHEPIR-SRVKXCTJSA-N 0.000 description 1
- KPYAOIVPJKPIOU-KKUMJFAQSA-N Leu-Lys-Lys Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCCCN)C(O)=O KPYAOIVPJKPIOU-KKUMJFAQSA-N 0.000 description 1
- VVQJGYPTIYOFBR-IHRRRGAJSA-N Leu-Lys-Met Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCSC)C(=O)O)N VVQJGYPTIYOFBR-IHRRRGAJSA-N 0.000 description 1
- WMIOEVKKYIMVKI-DCAQKATOSA-N Leu-Pro-Ala Chemical compound [H]N[C@@H](CC(C)C)C(=O)N1CCC[C@H]1C(=O)N[C@@H](C)C(O)=O WMIOEVKKYIMVKI-DCAQKATOSA-N 0.000 description 1
- JDBQSGMJBMPNFT-AVGNSLFASA-N Leu-Pro-Val Chemical compound CC(C)C[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](C(C)C)C(O)=O JDBQSGMJBMPNFT-AVGNSLFASA-N 0.000 description 1
- KIZIOFNVSOSKJI-CIUDSAMLSA-N Leu-Ser-Cys Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CS)C(=O)O)N KIZIOFNVSOSKJI-CIUDSAMLSA-N 0.000 description 1
- XOWMDXHFSBCAKQ-SRVKXCTJSA-N Leu-Ser-Leu Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CO)C(=O)N[C@H](C(O)=O)CC(C)C XOWMDXHFSBCAKQ-SRVKXCTJSA-N 0.000 description 1
- BRTVHXHCUSXYRI-CIUDSAMLSA-N Leu-Ser-Ser Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CO)C(=O)N[C@@H](CO)C(O)=O BRTVHXHCUSXYRI-CIUDSAMLSA-N 0.000 description 1
- SVBJIZVVYJYGLA-DCAQKATOSA-N Leu-Ser-Val Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(O)=O SVBJIZVVYJYGLA-DCAQKATOSA-N 0.000 description 1
- ZJZNLRVCZWUONM-JXUBOQSCSA-N Leu-Thr-Ala Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C)C(O)=O ZJZNLRVCZWUONM-JXUBOQSCSA-N 0.000 description 1
- LFSQWRSVPNKJGP-WDCWCFNPSA-N Leu-Thr-Glu Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@H](C(O)=O)CCC(O)=O LFSQWRSVPNKJGP-WDCWCFNPSA-N 0.000 description 1
- VHTIZYYHIUHMCA-JYJNAYRXSA-N Leu-Tyr-Gln Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CCC(N)=O)C(O)=O VHTIZYYHIUHMCA-JYJNAYRXSA-N 0.000 description 1
- MVJRBCJCRYGCKV-GVXVVHGQSA-N Leu-Val-Gln Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(N)=O)C(O)=O MVJRBCJCRYGCKV-GVXVVHGQSA-N 0.000 description 1
- QESXLSQLQHHTIX-RHYQMDGZSA-N Leu-Val-Thr Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O QESXLSQLQHHTIX-RHYQMDGZSA-N 0.000 description 1
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 description 1
- 239000012097 Lipofectamine 2000 Substances 0.000 description 1
- 101710099301 Low affinity immunoglobulin gamma Fc region receptor III-A Proteins 0.000 description 1
- XFIHDSBIPWEYJJ-YUMQZZPRSA-N Lys-Ala-Gly Chemical compound OC(=O)CNC(=O)[C@H](C)NC(=O)[C@@H](N)CCCCN XFIHDSBIPWEYJJ-YUMQZZPRSA-N 0.000 description 1
- KNKHAVVBVXKOGX-JXUBOQSCSA-N Lys-Ala-Thr Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O KNKHAVVBVXKOGX-JXUBOQSCSA-N 0.000 description 1
- GAOJCVKPIGHTGO-UWVGGRQHSA-N Lys-Arg-Gly Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CCCNC(N)=N)C(=O)NCC(O)=O GAOJCVKPIGHTGO-UWVGGRQHSA-N 0.000 description 1
- NQCJGQHHYZNUDK-DCAQKATOSA-N Lys-Arg-Ser Chemical compound NCCCC[C@H](N)C(=O)N[C@H](C(=O)N[C@@H](CO)C(O)=O)CCCN=C(N)N NQCJGQHHYZNUDK-DCAQKATOSA-N 0.000 description 1
- NCTDKZKNBDZDOL-GARJFASQSA-N Lys-Asn-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CC(=O)N)NC(=O)[C@H](CCCCN)N)C(=O)O NCTDKZKNBDZDOL-GARJFASQSA-N 0.000 description 1
- KWUKZRFFKPLUPE-HJGDQZAQSA-N Lys-Asp-Thr Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O KWUKZRFFKPLUPE-HJGDQZAQSA-N 0.000 description 1
- AIPHUKOBUXJNKM-KKUMJFAQSA-N Lys-Cys-Phe Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CS)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O AIPHUKOBUXJNKM-KKUMJFAQSA-N 0.000 description 1
- MQMIRLVJXQNTRJ-SDDRHHMPSA-N Lys-Gln-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CCCCN)N)C(=O)O MQMIRLVJXQNTRJ-SDDRHHMPSA-N 0.000 description 1
- HEWWNLVEWBJBKA-WDCWCFNPSA-N Lys-Gln-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)[C@H](CCC(N)=O)NC(=O)[C@@H](N)CCCCN HEWWNLVEWBJBKA-WDCWCFNPSA-N 0.000 description 1
- CRNNMTHBMRFQNG-GUBZILKMSA-N Lys-Glu-Cys Chemical compound C(CCN)C[C@@H](C(=O)N[C@@H](CCC(=O)O)C(=O)N[C@@H](CS)C(=O)O)N CRNNMTHBMRFQNG-GUBZILKMSA-N 0.000 description 1
- ONPDTSFZAIWMDI-AVGNSLFASA-N Lys-Leu-Gln Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(N)=O)C(O)=O ONPDTSFZAIWMDI-AVGNSLFASA-N 0.000 description 1
- SKRGVGLIRUGANF-AVGNSLFASA-N Lys-Leu-Glu Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(O)=O)C(O)=O SKRGVGLIRUGANF-AVGNSLFASA-N 0.000 description 1
- AIRZWUMAHCDDHR-KKUMJFAQSA-N Lys-Leu-Leu Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC(C)C)C(O)=O AIRZWUMAHCDDHR-KKUMJFAQSA-N 0.000 description 1
- URGPVYGVWLIRGT-DCAQKATOSA-N Lys-Met-Ala Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CCSC)C(=O)N[C@@H](C)C(O)=O URGPVYGVWLIRGT-DCAQKATOSA-N 0.000 description 1
- JYVCOTWSRGFABJ-DCAQKATOSA-N Lys-Met-Ser Chemical compound CSCC[C@@H](C(=O)N[C@@H](CO)C(=O)O)NC(=O)[C@H](CCCCN)N JYVCOTWSRGFABJ-DCAQKATOSA-N 0.000 description 1
- PDIDTSZKKFEDMB-UWVGGRQHSA-N Lys-Pro-Gly Chemical compound [H]N[C@@H](CCCCN)C(=O)N1CCC[C@H]1C(=O)NCC(O)=O PDIDTSZKKFEDMB-UWVGGRQHSA-N 0.000 description 1
- WQDKIVRHTQYJSN-DCAQKATOSA-N Lys-Ser-Arg Chemical compound C(CCN)C[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CCCN=C(N)N)C(=O)O)N WQDKIVRHTQYJSN-DCAQKATOSA-N 0.000 description 1
- DNWBUCHHMRQWCZ-GUBZILKMSA-N Lys-Ser-Gln Chemical compound NCCCC[C@H](N)C(=O)N[C@@H](CO)C(=O)N[C@H](C(O)=O)CCC(N)=O DNWBUCHHMRQWCZ-GUBZILKMSA-N 0.000 description 1
- DIBZLYZXTSVGLN-CIUDSAMLSA-N Lys-Ser-Ser Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CO)C(=O)N[C@@H](CO)C(O)=O DIBZLYZXTSVGLN-CIUDSAMLSA-N 0.000 description 1
- QVTDVTONTRSQMF-WDCWCFNPSA-N Lys-Thr-Glu Chemical compound OC(=O)CC[C@@H](C(O)=O)NC(=O)[C@H]([C@H](O)C)NC(=O)[C@@H](N)CCCCN QVTDVTONTRSQMF-WDCWCFNPSA-N 0.000 description 1
- MIMXMVDLMDMOJD-BZSNNMDCSA-N Lys-Tyr-Leu Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(C)C)C(O)=O MIMXMVDLMDMOJD-BZSNNMDCSA-N 0.000 description 1
- GILLQRYAWOMHED-DCAQKATOSA-N Lys-Val-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@H](C(C)C)NC(=O)[C@@H](N)CCCCN GILLQRYAWOMHED-DCAQKATOSA-N 0.000 description 1
- RIPJMCFGQHGHNP-RHYQMDGZSA-N Lys-Val-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](C(C)C)NC(=O)[C@H](CCCCN)N)O RIPJMCFGQHGHNP-RHYQMDGZSA-N 0.000 description 1
- 102000043129 MHC class I family Human genes 0.000 description 1
- 108091054437 MHC class I family Proteins 0.000 description 1
- 206010025598 Malignant hydatidiform mole Diseases 0.000 description 1
- 108010052285 Membrane Proteins Proteins 0.000 description 1
- BXNZDLVLGYYFIB-FXQIFTODSA-N Met-Asn-Cys Chemical compound CSCC[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H](CS)C(=O)O)N BXNZDLVLGYYFIB-FXQIFTODSA-N 0.000 description 1
- CAODKDAPYGUMLK-FXQIFTODSA-N Met-Asn-Ser Chemical compound CSCC[C@H](N)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CO)C(O)=O CAODKDAPYGUMLK-FXQIFTODSA-N 0.000 description 1
- QWTGQXGNNMIUCW-BPUTZDHNSA-N Met-Asn-Trp Chemical compound [H]N[C@@H](CCSC)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(O)=O QWTGQXGNNMIUCW-BPUTZDHNSA-N 0.000 description 1
- VOOINLQYUZOREH-SRVKXCTJSA-N Met-Gln-Leu Chemical compound CC(C)C[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CCSC)N VOOINLQYUZOREH-SRVKXCTJSA-N 0.000 description 1
- ABHVWYPPHDYFNY-WDSOQIARSA-N Met-His-Trp Chemical compound C([C@H](NC(=O)[C@@H](N)CCSC)C(=O)N[C@@H](CC=1C2=CC=CC=C2NC=1)C(O)=O)C1=CN=CN1 ABHVWYPPHDYFNY-WDSOQIARSA-N 0.000 description 1
- JHDNAOVJJQSMMM-GMOBBJLQSA-N Met-Ile-Asp Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC(=O)O)C(=O)O)NC(=O)[C@H](CCSC)N JHDNAOVJJQSMMM-GMOBBJLQSA-N 0.000 description 1
- AXHNAGAYRGCDLG-UWVGGRQHSA-N Met-Lys-Gly Chemical compound CSCC[C@H](N)C(=O)N[C@@H](CCCCN)C(=O)NCC(O)=O AXHNAGAYRGCDLG-UWVGGRQHSA-N 0.000 description 1
- RSOMVHWMIAZNLE-HJWJTTGWSA-N Met-Phe-Ile Chemical compound [H]N[C@@H](CCSC)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O RSOMVHWMIAZNLE-HJWJTTGWSA-N 0.000 description 1
- GGXZOTSDJJTDGB-GUBZILKMSA-N Met-Ser-Val Chemical compound [H]N[C@@H](CCSC)C(=O)N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(O)=O GGXZOTSDJJTDGB-GUBZILKMSA-N 0.000 description 1
- WSPQHZOMTFFWGH-XGEHTFHBSA-N Met-Thr-Cys Chemical compound CSCC[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CS)C(O)=O WSPQHZOMTFFWGH-XGEHTFHBSA-N 0.000 description 1
- SPSSJSICDYYTQN-HJGDQZAQSA-N Met-Thr-Gln Chemical compound CSCC[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@H](C(O)=O)CCC(N)=O SPSSJSICDYYTQN-HJGDQZAQSA-N 0.000 description 1
- 102000003792 Metallothionein Human genes 0.000 description 1
- 108090000157 Metallothionein Proteins 0.000 description 1
- 206010027476 Metastases Diseases 0.000 description 1
- 241001465754 Metazoa Species 0.000 description 1
- 241000713333 Mouse mammary tumor virus Species 0.000 description 1
- 101710135898 Myc proto-oncogene protein Proteins 0.000 description 1
- 102100038895 Myc proto-oncogene protein Human genes 0.000 description 1
- 201000007224 Myeloproliferative neoplasm Diseases 0.000 description 1
- 102000003505 Myosin Human genes 0.000 description 1
- 108060008487 Myosin Proteins 0.000 description 1
- SITLTJHOQZFJGG-UHFFFAOYSA-N N-L-alpha-glutamyl-L-valine Natural products CC(C)C(C(O)=O)NC(=O)C(N)CCC(O)=O SITLTJHOQZFJGG-UHFFFAOYSA-N 0.000 description 1
- PESQCPHRXOFIPX-UHFFFAOYSA-N N-L-methionyl-L-tyrosine Natural products CSCCC(N)C(=O)NC(C(O)=O)CC1=CC=C(O)C=C1 PESQCPHRXOFIPX-UHFFFAOYSA-N 0.000 description 1
- XMBSYZWANAQXEV-UHFFFAOYSA-N N-alpha-L-glutamyl-L-phenylalanine Natural products OC(=O)CCC(N)C(=O)NC(C(O)=O)CC1=CC=CC=C1 XMBSYZWANAQXEV-UHFFFAOYSA-N 0.000 description 1
- KZNQNBZMBZJQJO-UHFFFAOYSA-N N-glycyl-L-proline Natural products NCC(=O)N1CCCC1C(O)=O KZNQNBZMBZJQJO-UHFFFAOYSA-N 0.000 description 1
- 108010079364 N-glycylalanine Proteins 0.000 description 1
- 108010066427 N-valyltryptophan Proteins 0.000 description 1
- 208000034176 Neoplasms, Germ Cell and Embryonal Diseases 0.000 description 1
- 208000015914 Non-Hodgkin lymphomas Diseases 0.000 description 1
- 241000283973 Oryctolagus cuniculus Species 0.000 description 1
- 208000007571 Ovarian Epithelial Carcinoma Diseases 0.000 description 1
- 240000007019 Oxalis corniculata Species 0.000 description 1
- MDHZEOMXGNBSIL-DLOVCJGASA-N Phe-Ala-Cys Chemical compound C[C@@H](C(=O)N[C@@H](CS)C(=O)O)NC(=O)[C@H](CC1=CC=CC=C1)N MDHZEOMXGNBSIL-DLOVCJGASA-N 0.000 description 1
- NOFBJKKOPKJDCO-KKXDTOCCSA-N Phe-Ala-Tyr Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O NOFBJKKOPKJDCO-KKXDTOCCSA-N 0.000 description 1
- JIYJYFIXQTYDNF-YDHLFZDLSA-N Phe-Asn-Val Chemical compound CC(C)[C@@H](C(=O)O)NC(=O)[C@H](CC(=O)N)NC(=O)[C@H](CC1=CC=CC=C1)N JIYJYFIXQTYDNF-YDHLFZDLSA-N 0.000 description 1
- LLGTYVHITPVGKR-RYUDHWBXSA-N Phe-Gln-Gly Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CCC(N)=O)C(=O)NCC(O)=O LLGTYVHITPVGKR-RYUDHWBXSA-N 0.000 description 1
- YYKZDTVQHTUKDW-RYUDHWBXSA-N Phe-Gly-Gln Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)NCC(=O)N[C@@H](CCC(=O)N)C(=O)O)N YYKZDTVQHTUKDW-RYUDHWBXSA-N 0.000 description 1
- NAXPHWZXEXNDIW-JTQLQIEISA-N Phe-Gly-Gly Chemical compound OC(=O)CNC(=O)CNC(=O)[C@@H](N)CC1=CC=CC=C1 NAXPHWZXEXNDIW-JTQLQIEISA-N 0.000 description 1
- RSPUIENXSJYZQO-JYJNAYRXSA-N Phe-Leu-Gln Chemical compound NC(=O)CC[C@@H](C(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@@H](N)CC1=CC=CC=C1 RSPUIENXSJYZQO-JYJNAYRXSA-N 0.000 description 1
- AUJWXNGCAQWLEI-KBPBESRZSA-N Phe-Lys-Gly Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CCCCN)C(=O)NCC(O)=O AUJWXNGCAQWLEI-KBPBESRZSA-N 0.000 description 1
- IEOHQGFKHXUALJ-JYJNAYRXSA-N Phe-Met-Arg Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CCSC)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O IEOHQGFKHXUALJ-JYJNAYRXSA-N 0.000 description 1
- GNRMAQSIROFNMI-IXOXFDKPSA-N Phe-Thr-Ser Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CO)C(O)=O GNRMAQSIROFNMI-IXOXFDKPSA-N 0.000 description 1
- 102000004160 Phosphoric Monoester Hydrolases Human genes 0.000 description 1
- 108090000608 Phosphoric Monoester Hydrolases Proteins 0.000 description 1
- 108091000080 Phosphotransferase Proteins 0.000 description 1
- 241000709664 Picornaviridae Species 0.000 description 1
- 108010039918 Polylysine Proteins 0.000 description 1
- 241001672814 Porcine teschovirus 1 Species 0.000 description 1
- 241000288906 Primates Species 0.000 description 1
- FZHBZMDRDASUHN-NAKRPEOUSA-N Pro-Ala-Ile Chemical compound CC[C@H](C)[C@H](NC(=O)[C@H](C)NC(=O)[C@@H]1CCCN1)C(O)=O FZHBZMDRDASUHN-NAKRPEOUSA-N 0.000 description 1
- BNBBNGZZKQUWCD-IUCAKERBSA-N Pro-Arg-Gly Chemical compound NC(N)=NCCC[C@@H](C(=O)NCC(O)=O)NC(=O)[C@@H]1CCCN1 BNBBNGZZKQUWCD-IUCAKERBSA-N 0.000 description 1
- QSKCKTUQPICLSO-AVGNSLFASA-N Pro-Arg-Lys Chemical compound C1C[C@H](NC1)C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CCCCN)C(=O)O QSKCKTUQPICLSO-AVGNSLFASA-N 0.000 description 1
- ICTZKEXYDDZZFP-SRVKXCTJSA-N Pro-Arg-Pro Chemical compound N([C@@H](CCCN=C(N)N)C(=O)N1[C@@H](CCC1)C(O)=O)C(=O)[C@@H]1CCCN1 ICTZKEXYDDZZFP-SRVKXCTJSA-N 0.000 description 1
- SGCZFWSQERRKBD-BQBZGAKWSA-N Pro-Asp-Gly Chemical compound OC(=O)CNC(=O)[C@H](CC(O)=O)NC(=O)[C@@H]1CCCN1 SGCZFWSQERRKBD-BQBZGAKWSA-N 0.000 description 1
- GDXZRWYXJSGWIV-GMOBBJLQSA-N Pro-Asp-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H](CC(=O)O)NC(=O)[C@@H]1CCCN1 GDXZRWYXJSGWIV-GMOBBJLQSA-N 0.000 description 1
- ZCXQTRXYZOSGJR-FXQIFTODSA-N Pro-Asp-Ser Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CO)C(O)=O ZCXQTRXYZOSGJR-FXQIFTODSA-N 0.000 description 1
- DEDANIDYQAPTFI-IHRRRGAJSA-N Pro-Asp-Tyr Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O DEDANIDYQAPTFI-IHRRRGAJSA-N 0.000 description 1
- LHALYDBUDCWMDY-CIUDSAMLSA-N Pro-Glu-Ala Chemical compound C[C@H](NC(=O)[C@H](CCC(O)=O)NC(=O)[C@@H]1CCCN1)C(O)=O LHALYDBUDCWMDY-CIUDSAMLSA-N 0.000 description 1
- VPEVBAUSTBWQHN-NHCYSSNCSA-N Pro-Glu-Val Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](C(C)C)C(O)=O VPEVBAUSTBWQHN-NHCYSSNCSA-N 0.000 description 1
- DMKWYMWNEKIPFC-IUCAKERBSA-N Pro-Gly-Arg Chemical compound [H]N1CCC[C@H]1C(=O)NCC(=O)N[C@@H](CCCNC(N)=N)C(O)=O DMKWYMWNEKIPFC-IUCAKERBSA-N 0.000 description 1
- ULIWFCCJIOEHMU-BQBZGAKWSA-N Pro-Gly-Asp Chemical compound OC(=O)C[C@@H](C(O)=O)NC(=O)CNC(=O)[C@@H]1CCCN1 ULIWFCCJIOEHMU-BQBZGAKWSA-N 0.000 description 1
- JMVQDLDPDBXAAX-YUMQZZPRSA-N Pro-Gly-Gln Chemical compound NC(=O)CC[C@@H](C(O)=O)NC(=O)CNC(=O)[C@@H]1CCCN1 JMVQDLDPDBXAAX-YUMQZZPRSA-N 0.000 description 1
- VYWNORHENYEQDW-YUMQZZPRSA-N Pro-Gly-Glu Chemical compound OC(=O)CC[C@@H](C(O)=O)NC(=O)CNC(=O)[C@@H]1CCCN1 VYWNORHENYEQDW-YUMQZZPRSA-N 0.000 description 1
- FKLSMYYLJHYPHH-UWVGGRQHSA-N Pro-Gly-Leu Chemical compound [H]N1CCC[C@H]1C(=O)NCC(=O)N[C@@H](CC(C)C)C(O)=O FKLSMYYLJHYPHH-UWVGGRQHSA-N 0.000 description 1
- ULWBBFKQBDNGOY-RWMBFGLXSA-N Pro-Lys-Pro Chemical compound C1C[C@H](NC1)C(=O)N[C@@H](CCCCN)C(=O)N2CCC[C@@H]2C(=O)O ULWBBFKQBDNGOY-RWMBFGLXSA-N 0.000 description 1
- KDBHVPXBQADZKY-GUBZILKMSA-N Pro-Pro-Ala Chemical compound OC(=O)[C@H](C)NC(=O)[C@@H]1CCCN1C(=O)[C@H]1NCCC1 KDBHVPXBQADZKY-GUBZILKMSA-N 0.000 description 1
- SBVPYBFMIGDIDX-SRVKXCTJSA-N Pro-Pro-Pro Chemical compound OC(=O)[C@@H]1CCCN1C(=O)[C@H]1N(C(=O)[C@H]2NCCC2)CCC1 SBVPYBFMIGDIDX-SRVKXCTJSA-N 0.000 description 1
- JDJMFMVVJHLWDP-UNQGMJICSA-N Pro-Thr-Phe Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O JDJMFMVVJHLWDP-UNQGMJICSA-N 0.000 description 1
- 229940124158 Protease/peptidase inhibitor Drugs 0.000 description 1
- 239000012083 RIPA buffer Substances 0.000 description 1
- 230000010799 Receptor Interactions Effects 0.000 description 1
- 108020005091 Replication Origin Proteins 0.000 description 1
- 108091027981 Response element Proteins 0.000 description 1
- 241000283984 Rodentia Species 0.000 description 1
- 241000714474 Rous sarcoma virus Species 0.000 description 1
- 206010039491 Sarcoma Diseases 0.000 description 1
- HRNQLKCLPVKZNE-CIUDSAMLSA-N Ser-Ala-Leu Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](C)C(=O)N[C@@H](CC(C)C)C(O)=O HRNQLKCLPVKZNE-CIUDSAMLSA-N 0.000 description 1
- YQHZVYJAGWMHES-ZLUOBGJFSA-N Ser-Ala-Ser Chemical compound OC[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@@H](CO)C(O)=O YQHZVYJAGWMHES-ZLUOBGJFSA-N 0.000 description 1
- NLQUOHDCLSFABG-GUBZILKMSA-N Ser-Arg-Arg Chemical compound NC(N)=NCCC[C@H](NC(=O)[C@H](CO)N)C(=O)N[C@@H](CCCN=C(N)N)C(O)=O NLQUOHDCLSFABG-GUBZILKMSA-N 0.000 description 1
- YUSRGTQIPCJNHQ-CIUDSAMLSA-N Ser-Arg-Glu Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(O)=O)C(O)=O YUSRGTQIPCJNHQ-CIUDSAMLSA-N 0.000 description 1
- WDXYVIIVDIDOSX-DCAQKATOSA-N Ser-Arg-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)[C@@H](N)CO)CCCN=C(N)N WDXYVIIVDIDOSX-DCAQKATOSA-N 0.000 description 1
- QVOGDCQNGLBNCR-FXQIFTODSA-N Ser-Arg-Ser Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CO)C(O)=O QVOGDCQNGLBNCR-FXQIFTODSA-N 0.000 description 1
- HBOABDXGTMMDSE-GUBZILKMSA-N Ser-Arg-Val Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](C(C)C)C(O)=O HBOABDXGTMMDSE-GUBZILKMSA-N 0.000 description 1
- FIDMVVBUOCMMJG-CIUDSAMLSA-N Ser-Asn-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@@H](N)CO FIDMVVBUOCMMJG-CIUDSAMLSA-N 0.000 description 1
- SWSRFJZZMNLMLY-ZKWXMUAHSA-N Ser-Asp-Val Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](C(C)C)C(O)=O SWSRFJZZMNLMLY-ZKWXMUAHSA-N 0.000 description 1
- BLPYXIXXCFVIIF-FXQIFTODSA-N Ser-Cys-Arg Chemical compound C(C[C@@H](C(=O)O)NC(=O)[C@H](CS)NC(=O)[C@H](CO)N)CN=C(N)N BLPYXIXXCFVIIF-FXQIFTODSA-N 0.000 description 1
- ULVMNZOKDBHKKI-ACZMJKKPSA-N Ser-Gln-Asp Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(O)=O)C(O)=O ULVMNZOKDBHKKI-ACZMJKKPSA-N 0.000 description 1
- GWMXFEMMBHOKDX-AVGNSLFASA-N Ser-Gln-Phe Chemical compound OC[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 GWMXFEMMBHOKDX-AVGNSLFASA-N 0.000 description 1
- GZBKRJVCRMZAST-XKBZYTNZSA-N Ser-Glu-Thr Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O GZBKRJVCRMZAST-XKBZYTNZSA-N 0.000 description 1
- OHKFXGKHSJKKAL-NRPADANISA-N Ser-Glu-Val Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](C(C)C)C(O)=O OHKFXGKHSJKKAL-NRPADANISA-N 0.000 description 1
- UQFYNFTYDHUIMI-WHFBIAKZSA-N Ser-Gly-Ala Chemical compound OC(=O)[C@H](C)NC(=O)CNC(=O)[C@@H](N)CO UQFYNFTYDHUIMI-WHFBIAKZSA-N 0.000 description 1
- MUARUIBTKQJKFY-WHFBIAKZSA-N Ser-Gly-Asp Chemical compound [H]N[C@@H](CO)C(=O)NCC(=O)N[C@@H](CC(O)=O)C(O)=O MUARUIBTKQJKFY-WHFBIAKZSA-N 0.000 description 1
- SVWQEIRZHHNBIO-WHFBIAKZSA-N Ser-Gly-Cys Chemical compound [H]N[C@@H](CO)C(=O)NCC(=O)N[C@@H](CS)C(O)=O SVWQEIRZHHNBIO-WHFBIAKZSA-N 0.000 description 1
- IOVHBRCQOGWAQH-ZKWXMUAHSA-N Ser-Gly-Ile Chemical compound [H]N[C@@H](CO)C(=O)NCC(=O)N[C@@H]([C@@H](C)CC)C(O)=O IOVHBRCQOGWAQH-ZKWXMUAHSA-N 0.000 description 1
- FUMGHWDRRFCKEP-CIUDSAMLSA-N Ser-Leu-Ala Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](C)C(O)=O FUMGHWDRRFCKEP-CIUDSAMLSA-N 0.000 description 1
- ZIFYDQAFEMIZII-GUBZILKMSA-N Ser-Leu-Glu Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(O)=O)C(O)=O ZIFYDQAFEMIZII-GUBZILKMSA-N 0.000 description 1
- VMLONWHIORGALA-SRVKXCTJSA-N Ser-Leu-Leu Chemical compound CC(C)C[C@@H](C([O-])=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@@H]([NH3+])CO VMLONWHIORGALA-SRVKXCTJSA-N 0.000 description 1
- KCGIREHVWRXNDH-GARJFASQSA-N Ser-Leu-Pro Chemical compound CC(C)C[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CO)N KCGIREHVWRXNDH-GARJFASQSA-N 0.000 description 1
- MUJQWSAWLLRJCE-KATARQTJSA-N Ser-Leu-Thr Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O MUJQWSAWLLRJCE-KATARQTJSA-N 0.000 description 1
- JWOBLHJRDADHLN-KKUMJFAQSA-N Ser-Leu-Tyr Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O JWOBLHJRDADHLN-KKUMJFAQSA-N 0.000 description 1
- HDBOEVPDIDDEPC-CIUDSAMLSA-N Ser-Lys-Asn Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(N)=O)C(O)=O HDBOEVPDIDDEPC-CIUDSAMLSA-N 0.000 description 1
- QMCDMHWAKMUGJE-IHRRRGAJSA-N Ser-Phe-Val Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](C(C)C)C(O)=O QMCDMHWAKMUGJE-IHRRRGAJSA-N 0.000 description 1
- XZKQVQKUZMAADP-IMJSIDKUSA-N Ser-Ser Chemical compound OC[C@H](N)C(=O)N[C@@H](CO)C(O)=O XZKQVQKUZMAADP-IMJSIDKUSA-N 0.000 description 1
- BMKNXTJLHFIAAH-CIUDSAMLSA-N Ser-Ser-Leu Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(O)=O BMKNXTJLHFIAAH-CIUDSAMLSA-N 0.000 description 1
- ATEQEHCGZKBEMU-GQGQLFGLSA-N Ser-Trp-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H](CC1=CNC2=CC=CC=C21)NC(=O)[C@H](CO)N ATEQEHCGZKBEMU-GQGQLFGLSA-N 0.000 description 1
- PIQRHJQWEPWFJG-UWJYBYFXSA-N Ser-Tyr-Ala Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](C)C(O)=O PIQRHJQWEPWFJG-UWJYBYFXSA-N 0.000 description 1
- FGBLCMLXHRPVOF-IHRRRGAJSA-N Ser-Tyr-Arg Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O FGBLCMLXHRPVOF-IHRRRGAJSA-N 0.000 description 1
- PQEQXWRVHQAAKS-SRVKXCTJSA-N Ser-Tyr-Asn Chemical compound NC(=O)C[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)[C@H](CO)N)CC1=CC=C(O)C=C1 PQEQXWRVHQAAKS-SRVKXCTJSA-N 0.000 description 1
- VVKVHAOOUGNDPJ-SRVKXCTJSA-N Ser-Tyr-Ser Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CO)C(O)=O VVKVHAOOUGNDPJ-SRVKXCTJSA-N 0.000 description 1
- JZRYFUGREMECBH-XPUUQOCRSA-N Ser-Val-Gly Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(=O)NCC(O)=O JZRYFUGREMECBH-XPUUQOCRSA-N 0.000 description 1
- 206010041067 Small cell lung cancer Diseases 0.000 description 1
- 241001493546 Suina Species 0.000 description 1
- 230000024932 T cell mediated immunity Effects 0.000 description 1
- 102100027222 T-lymphocyte activation antigen CD80 Human genes 0.000 description 1
- 206010043276 Teratoma Diseases 0.000 description 1
- 241001648840 Thosea asigna virus Species 0.000 description 1
- BSNZTJXVDOINSR-JXUBOQSCSA-N Thr-Ala-Leu Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C)C(=O)N[C@@H](CC(C)C)C(O)=O BSNZTJXVDOINSR-JXUBOQSCSA-N 0.000 description 1
- GFDUZZACIWNMPE-KZVJFYERSA-N Thr-Ala-Met Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C)C(=O)N[C@@H](CCSC)C(O)=O GFDUZZACIWNMPE-KZVJFYERSA-N 0.000 description 1
- CAJFZCICSVBOJK-SHGPDSBTSA-N Thr-Ala-Thr Chemical compound C[C@@H](O)[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O CAJFZCICSVBOJK-SHGPDSBTSA-N 0.000 description 1
- XSLXHSYIVPGEER-KZVJFYERSA-N Thr-Ala-Val Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C)C(=O)N[C@@H](C(C)C)C(O)=O XSLXHSYIVPGEER-KZVJFYERSA-N 0.000 description 1
- TWLMXDWFVNEFFK-FJXKBIBVSA-N Thr-Arg-Gly Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)NCC(O)=O TWLMXDWFVNEFFK-FJXKBIBVSA-N 0.000 description 1
- JBHMLZSKIXMVFS-XVSYOHENSA-N Thr-Asn-Phe Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O JBHMLZSKIXMVFS-XVSYOHENSA-N 0.000 description 1
- LMMDEZPNUTZJAY-GCJQMDKQSA-N Thr-Asp-Ala Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](C)C(O)=O LMMDEZPNUTZJAY-GCJQMDKQSA-N 0.000 description 1
- KRPKYGOFYUNIGM-XVSYOHENSA-N Thr-Asp-Phe Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CC(=O)O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)O)N)O KRPKYGOFYUNIGM-XVSYOHENSA-N 0.000 description 1
- DCLBXIWHLVEPMQ-JRQIVUDYSA-N Thr-Asp-Tyr Chemical compound C[C@@H](O)[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 DCLBXIWHLVEPMQ-JRQIVUDYSA-N 0.000 description 1
- UZJDBCHMIQXLOQ-HEIBUPTGSA-N Thr-Cys-Thr Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CS)C(=O)N[C@@H]([C@@H](C)O)C(=O)O)N)O UZJDBCHMIQXLOQ-HEIBUPTGSA-N 0.000 description 1
- LIXBDERDAGNVAV-XKBZYTNZSA-N Thr-Gln-Ser Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CO)C(O)=O LIXBDERDAGNVAV-XKBZYTNZSA-N 0.000 description 1
- KGKWKSSSQGGYAU-SUSMZKCASA-N Thr-Gln-Thr Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)N[C@@H]([C@@H](C)O)C(=O)O)N)O KGKWKSSSQGGYAU-SUSMZKCASA-N 0.000 description 1
- SLUWOCTZVGMURC-BFHQHQDPSA-N Thr-Gly-Ala Chemical compound C[C@@H](O)[C@H](N)C(=O)NCC(=O)N[C@@H](C)C(O)=O SLUWOCTZVGMURC-BFHQHQDPSA-N 0.000 description 1
- GXUWHVZYDAHFSV-FLBSBUHZSA-N Thr-Ile-Thr Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H]([C@@H](C)O)C(O)=O GXUWHVZYDAHFSV-FLBSBUHZSA-N 0.000 description 1
- RFKVQLIXNVEOMB-WEDXCCLWSA-N Thr-Leu-Gly Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CC(C)C)C(=O)NCC(=O)O)N)O RFKVQLIXNVEOMB-WEDXCCLWSA-N 0.000 description 1
- NCXVJIQMWSGRHY-KXNHARMFSA-N Thr-Leu-Pro Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CC(C)C)C(=O)N1CCC[C@@H]1C(=O)O)N)O NCXVJIQMWSGRHY-KXNHARMFSA-N 0.000 description 1
- ZSPQUTWLWGWTPS-HJGDQZAQSA-N Thr-Lys-Asp Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(O)=O)C(O)=O ZSPQUTWLWGWTPS-HJGDQZAQSA-N 0.000 description 1
- BIBYEFRASCNLAA-CDMKHQONSA-N Thr-Phe-Gly Chemical compound C[C@@H](O)[C@H](N)C(=O)N[C@H](C(=O)NCC(O)=O)CC1=CC=CC=C1 BIBYEFRASCNLAA-CDMKHQONSA-N 0.000 description 1
- WNQJTLATMXYSEL-OEAJRASXSA-N Thr-Phe-Leu Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CC(C)C)C(O)=O WNQJTLATMXYSEL-OEAJRASXSA-N 0.000 description 1
- MXNAOGFNFNKUPD-JHYOHUSXSA-N Thr-Phe-Thr Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H]([C@@H](C)O)C(O)=O MXNAOGFNFNKUPD-JHYOHUSXSA-N 0.000 description 1
- KERCOYANYUPLHJ-XGEHTFHBSA-N Thr-Pro-Ser Chemical compound C[C@@H](O)[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CO)C(O)=O KERCOYANYUPLHJ-XGEHTFHBSA-N 0.000 description 1
- IEZVHOULSUULHD-XGEHTFHBSA-N Thr-Ser-Val Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(O)=O IEZVHOULSUULHD-XGEHTFHBSA-N 0.000 description 1
- ZESGVALRVJIVLZ-VFCFLDTKSA-N Thr-Thr-Pro Chemical compound C[C@H]([C@@H](C(=O)N[C@@H]([C@@H](C)O)C(=O)N1CCC[C@@H]1C(=O)O)N)O ZESGVALRVJIVLZ-VFCFLDTKSA-N 0.000 description 1
- NJGMALCNYAMYCB-JRQIVUDYSA-N Thr-Tyr-Asn Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(N)=O)C(O)=O NJGMALCNYAMYCB-JRQIVUDYSA-N 0.000 description 1
- XVHAUVJXBFGUPC-RPTUDFQQSA-N Thr-Tyr-Phe Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O XVHAUVJXBFGUPC-RPTUDFQQSA-N 0.000 description 1
- OGOYMQWIWHGTGH-KZVJFYERSA-N Thr-Val-Ala Chemical compound C[C@@H](O)[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](C)C(O)=O OGOYMQWIWHGTGH-KZVJFYERSA-N 0.000 description 1
- MNYNCKZAEIAONY-XGEHTFHBSA-N Thr-Val-Ser Chemical compound C[C@@H](O)[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CO)C(O)=O MNYNCKZAEIAONY-XGEHTFHBSA-N 0.000 description 1
- 101710150448 Transcriptional regulator Myc Proteins 0.000 description 1
- VZBWRZGNEPBRDE-HZUKXOBISA-N Trp-Ala-Pro Chemical compound C[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CC2=CNC3=CC=CC=C32)N VZBWRZGNEPBRDE-HZUKXOBISA-N 0.000 description 1
- AVYVKJMBNLPWRX-WFBYXXMGSA-N Trp-Ala-Ser Chemical compound C1=CC=C2C(C[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@@H](CO)C(O)=O)=CNC2=C1 AVYVKJMBNLPWRX-WFBYXXMGSA-N 0.000 description 1
- FEZASNVQLJQBHW-CABZTGNLSA-N Trp-Gly-Ala Chemical compound C1=CC=C2C(C[C@H](N)C(=O)NCC(=O)N[C@@H](C)C(O)=O)=CNC2=C1 FEZASNVQLJQBHW-CABZTGNLSA-N 0.000 description 1
- SVGAWGVHFIYAEE-JSGCOSHPSA-N Trp-Gly-Gln Chemical compound C1=CC=C2C(C[C@H](N)C(=O)NCC(=O)N[C@@H](CCC(N)=O)C(O)=O)=CNC2=C1 SVGAWGVHFIYAEE-JSGCOSHPSA-N 0.000 description 1
- KIMOCKLJBXHFIN-YLVFBTJISA-N Trp-Ile-Gly Chemical compound C1=CC=C2C(C[C@H](N)C(=O)N[C@@H]([C@@H](C)CC)C(=O)NCC(O)=O)=CNC2=C1 KIMOCKLJBXHFIN-YLVFBTJISA-N 0.000 description 1
- ZHDQRPWESGUDST-JBACZVJFSA-N Trp-Phe-Gln Chemical compound C([C@H](NC(=O)[C@H](CC=1C2=CC=CC=C2NC=1)N)C(=O)N[C@@H](CCC(N)=O)C(O)=O)C1=CC=CC=C1 ZHDQRPWESGUDST-JBACZVJFSA-N 0.000 description 1
- PKZIWSHDJYIPRH-JBACZVJFSA-N Trp-Tyr-Gln Chemical compound [H]N[C@@H](CC1=CNC2=C1C=CC=C2)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CCC(N)=O)C(O)=O PKZIWSHDJYIPRH-JBACZVJFSA-N 0.000 description 1
- XQMGDVVKFRLQKH-BBRMVZONSA-N Trp-Val-Gly Chemical compound C1=CC=C2C(C[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)NCC(O)=O)=CNC2=C1 XQMGDVVKFRLQKH-BBRMVZONSA-N 0.000 description 1
- QIVBCDIJIAJPQS-UHFFFAOYSA-N Tryptophan Natural products C1=CC=C2C(CC(N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-UHFFFAOYSA-N 0.000 description 1
- 108060008683 Tumor Necrosis Factor Receptor Proteins 0.000 description 1
- CDRYEAWHKJSGAF-BPNCWPANSA-N Tyr-Ala-Met Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](C)C(=O)N[C@@H](CCSC)C(O)=O CDRYEAWHKJSGAF-BPNCWPANSA-N 0.000 description 1
- IIJWXEUNETVJPV-IHRRRGAJSA-N Tyr-Arg-Ser Chemical compound C1=CC(=CC=C1C[C@@H](C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CO)C(=O)O)N)O IIJWXEUNETVJPV-IHRRRGAJSA-N 0.000 description 1
- OEVJGIHPQOXYFE-SRVKXCTJSA-N Tyr-Asn-Asp Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CC(O)=O)C(O)=O OEVJGIHPQOXYFE-SRVKXCTJSA-N 0.000 description 1
- MBFJIHUHHCJBSN-AVGNSLFASA-N Tyr-Asn-Gln Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CCC(N)=O)C(O)=O MBFJIHUHHCJBSN-AVGNSLFASA-N 0.000 description 1
- CTDPLKMBVALCGN-JSGCOSHPSA-N Tyr-Gly-Val Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)NCC(=O)N[C@@H](C(C)C)C(O)=O CTDPLKMBVALCGN-JSGCOSHPSA-N 0.000 description 1
- CVXURBLRELTJKO-BWAGICSOSA-N Tyr-His-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CC1=CN=CN1)NC(=O)[C@H](CC2=CC=C(C=C2)O)N)O CVXURBLRELTJKO-BWAGICSOSA-N 0.000 description 1
- DZKFGCNKEVMXFA-JUKXBJQTSA-N Tyr-Ile-His Chemical compound CC[C@H](C)[C@H](NC(=O)[C@@H](N)Cc1ccc(O)cc1)C(=O)N[C@@H](Cc1cnc[nH]1)C(O)=O DZKFGCNKEVMXFA-JUKXBJQTSA-N 0.000 description 1
- AZZLDIDWPZLCCW-ZEWNOJEFSA-N Tyr-Ile-Phe Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O AZZLDIDWPZLCCW-ZEWNOJEFSA-N 0.000 description 1
- LQGDFDYGDQEMGA-PXDAIIFMSA-N Tyr-Ile-Trp Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC1=CNC2=CC=CC=C21)C(=O)O)NC(=O)[C@H](CC3=CC=C(C=C3)O)N LQGDFDYGDQEMGA-PXDAIIFMSA-N 0.000 description 1
- MVFQLSPDMMFCMW-KKUMJFAQSA-N Tyr-Leu-Asn Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC(N)=O)C(O)=O MVFQLSPDMMFCMW-KKUMJFAQSA-N 0.000 description 1
- XDGPTBVOSHKDFT-KKUMJFAQSA-N Tyr-Met-Glu Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CCSC)C(=O)N[C@@H](CCC(O)=O)C(O)=O XDGPTBVOSHKDFT-KKUMJFAQSA-N 0.000 description 1
- UPODKYBYUBTWSV-BZSNNMDCSA-N Tyr-Phe-Cys Chemical compound C([C@H](N)C(=O)N[C@@H](CC=1C=CC=CC=1)C(=O)N[C@@H](CS)C(O)=O)C1=CC=C(O)C=C1 UPODKYBYUBTWSV-BZSNNMDCSA-N 0.000 description 1
- QFXVAFIHVWXXBJ-AVGNSLFASA-N Tyr-Ser-Glu Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CO)C(=O)N[C@@H](CCC(O)=O)C(O)=O QFXVAFIHVWXXBJ-AVGNSLFASA-N 0.000 description 1
- LUMQYLVYUIRHHU-YJRXYDGGSA-N Tyr-Ser-Thr Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CO)C(=O)N[C@@H]([C@@H](C)O)C(O)=O LUMQYLVYUIRHHU-YJRXYDGGSA-N 0.000 description 1
- LVFZXRQQQDTBQH-IRIUXVKKSA-N Tyr-Thr-Glu Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCC(O)=O)C(O)=O LVFZXRQQQDTBQH-IRIUXVKKSA-N 0.000 description 1
- LDKDSFQSEUOCOO-RPTUDFQQSA-N Tyr-Thr-Phe Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O LDKDSFQSEUOCOO-RPTUDFQQSA-N 0.000 description 1
- RMRFSFXLFWWAJZ-HJOGWXRNSA-N Tyr-Tyr-Tyr Chemical compound C([C@H](N)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(O)=O)C1=CC=C(O)C=C1 RMRFSFXLFWWAJZ-HJOGWXRNSA-N 0.000 description 1
- VKYDVKAKGDNZED-STECZYCISA-N Tyr-Val-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H](C(C)C)NC(=O)[C@H](CC1=CC=C(C=C1)O)N VKYDVKAKGDNZED-STECZYCISA-N 0.000 description 1
- JIODCDXKCJRMEH-NHCYSSNCSA-N Val-Arg-Gln Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CCC(=O)N)C(=O)O)N JIODCDXKCJRMEH-NHCYSSNCSA-N 0.000 description 1
- IVXJODPZRWHCCR-JYJNAYRXSA-N Val-Arg-Phe Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)O)N IVXJODPZRWHCCR-JYJNAYRXSA-N 0.000 description 1
- DBOXBUDEAJVKRE-LSJOCFKGSA-N Val-Asn-Val Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H](C(C)C)C(=O)O)N DBOXBUDEAJVKRE-LSJOCFKGSA-N 0.000 description 1
- NYTKXWLZSNRILS-IFFSRLJSSA-N Val-Gln-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](C(C)C)N)O NYTKXWLZSNRILS-IFFSRLJSSA-N 0.000 description 1
- OQWNEUXPKHIEJO-NRPADANISA-N Val-Glu-Ser Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCC(=O)O)C(=O)N[C@@H](CO)C(=O)O)N OQWNEUXPKHIEJO-NRPADANISA-N 0.000 description 1
- NXRAUQGGHPCJIB-RCOVLWMOSA-N Val-Gly-Asn Chemical compound CC(C)[C@H](N)C(=O)NCC(=O)N[C@@H](CC(N)=O)C(O)=O NXRAUQGGHPCJIB-RCOVLWMOSA-N 0.000 description 1
- HQYVQDRYODWONX-DCAQKATOSA-N Val-His-Ser Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)N[C@@H](CO)C(=O)O)N HQYVQDRYODWONX-DCAQKATOSA-N 0.000 description 1
- MYLNLEIZWHVENT-VKOGCVSHSA-N Val-Ile-Trp Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC1=CNC2=CC=CC=C21)C(=O)O)NC(=O)[C@H](C(C)C)N MYLNLEIZWHVENT-VKOGCVSHSA-N 0.000 description 1
- IEBGHUMBJXIXHM-AVGNSLFASA-N Val-Lys-Met Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCSC)C(=O)O)N IEBGHUMBJXIXHM-AVGNSLFASA-N 0.000 description 1
- HPANGHISDXDUQY-ULQDDVLXSA-N Val-Lys-Phe Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)O)N HPANGHISDXDUQY-ULQDDVLXSA-N 0.000 description 1
- CXWJFWAZIVWBOS-XQQFMLRXSA-N Val-Lys-Pro Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)N1CCC[C@@H]1C(=O)O)N CXWJFWAZIVWBOS-XQQFMLRXSA-N 0.000 description 1
- JAKHAONCJJZVHT-DCAQKATOSA-N Val-Lys-Ser Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CO)C(=O)O)N JAKHAONCJJZVHT-DCAQKATOSA-N 0.000 description 1
- HJSLDXZAZGFPDK-ULQDDVLXSA-N Val-Phe-Leu Chemical compound CC(C)C[C@@H](C(=O)O)NC(=O)[C@H](CC1=CC=CC=C1)NC(=O)[C@H](C(C)C)N HJSLDXZAZGFPDK-ULQDDVLXSA-N 0.000 description 1
- SSYBNWFXCFNRFN-GUBZILKMSA-N Val-Pro-Ser Chemical compound CC(C)[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CO)C(O)=O SSYBNWFXCFNRFN-GUBZILKMSA-N 0.000 description 1
- UGFMVXRXULGLNO-XPUUQOCRSA-N Val-Ser-Gly Chemical compound CC(C)[C@H](N)C(=O)N[C@@H](CO)C(=O)NCC(O)=O UGFMVXRXULGLNO-XPUUQOCRSA-N 0.000 description 1
- VHIZXDZMTDVFGX-DCAQKATOSA-N Val-Ser-Leu Chemical compound CC(C)C[C@@H](C(=O)O)NC(=O)[C@H](CO)NC(=O)[C@H](C(C)C)N VHIZXDZMTDVFGX-DCAQKATOSA-N 0.000 description 1
- UJMCYJKPDFQLHX-XGEHTFHBSA-N Val-Ser-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CO)NC(=O)[C@H](C(C)C)N)O UJMCYJKPDFQLHX-XGEHTFHBSA-N 0.000 description 1
- SDHZOOIGIUEPDY-JYJNAYRXSA-N Val-Ser-Trp Chemical compound C1=CC=C2C(C[C@H](NC(=O)[C@H](CO)NC(=O)[C@@H](N)C(C)C)C(O)=O)=CNC2=C1 SDHZOOIGIUEPDY-JYJNAYRXSA-N 0.000 description 1
- CEKSLIVSNNGOKH-KZVJFYERSA-N Val-Thr-Ala Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](C)C(=O)O)NC(=O)[C@H](C(C)C)N)O CEKSLIVSNNGOKH-KZVJFYERSA-N 0.000 description 1
- UVHFONIHVHLDDQ-IFFSRLJSSA-N Val-Thr-Glu Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CCC(=O)O)C(=O)O)NC(=O)[C@H](C(C)C)N)O UVHFONIHVHLDDQ-IFFSRLJSSA-N 0.000 description 1
- WUFHZIRMAZZWRS-OSUNSFLBSA-N Val-Thr-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H]([C@@H](C)O)NC(=O)[C@H](C(C)C)N WUFHZIRMAZZWRS-OSUNSFLBSA-N 0.000 description 1
- HTONZBWRYUKUKC-RCWTZXSCSA-N Val-Thr-Val Chemical compound CC(C)[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C(C)C)C(O)=O HTONZBWRYUKUKC-RCWTZXSCSA-N 0.000 description 1
- OWFGFHQMSBTKLX-UFYCRDLUSA-N Val-Tyr-Tyr Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)N[C@@H](CC2=CC=C(C=C2)O)C(=O)O)N OWFGFHQMSBTKLX-UFYCRDLUSA-N 0.000 description 1
- 241001416177 Vicugna pacos Species 0.000 description 1
- 208000012018 Yolk sac tumor Diseases 0.000 description 1
- QPMSXSBEVQLBIL-CZRHPSIPSA-N ac1mix0p Chemical compound C1=CC=C2N(C[C@H](C)CN(C)C)C3=CC(OC)=CC=C3SC2=C1.O([C@H]1[C@]2(OC)C=CC34C[C@@H]2[C@](C)(O)CCC)C2=C5[C@]41CCN(C)[C@@H]3CC5=CC=C2O QPMSXSBEVQLBIL-CZRHPSIPSA-N 0.000 description 1
- 208000024340 acute graft versus host disease Diseases 0.000 description 1
- 239000002671 adjuvant Substances 0.000 description 1
- 230000002411 adverse Effects 0.000 description 1
- 108010069020 alanyl-prolyl-glycine Proteins 0.000 description 1
- 108010005233 alanylglutamic acid Proteins 0.000 description 1
- 108010070944 alanylhistidine Proteins 0.000 description 1
- 108010070783 alanyltyrosine Proteins 0.000 description 1
- 238000011316 allogeneic transplantation Methods 0.000 description 1
- 230000001446 anti-myeloma Effects 0.000 description 1
- 230000006023 anti-tumor response Effects 0.000 description 1
- 210000000612 antigen-presenting cell Anatomy 0.000 description 1
- 239000002246 antineoplastic agent Substances 0.000 description 1
- 108010013835 arginine glutamate Proteins 0.000 description 1
- 108010001271 arginyl-glutamyl-arginine Proteins 0.000 description 1
- 108010038850 arginyl-isoleucyl-tyrosine Proteins 0.000 description 1
- 108010059459 arginyl-threonyl-phenylalanine Proteins 0.000 description 1
- 108010062796 arginyllysine Proteins 0.000 description 1
- 108010040443 aspartyl-aspartic acid Proteins 0.000 description 1
- 230000015572 biosynthetic process Effects 0.000 description 1
- 229960002685 biotin Drugs 0.000 description 1
- 235000020958 biotin Nutrition 0.000 description 1
- 239000011616 biotin Substances 0.000 description 1
- 238000009583 bone marrow aspiration Methods 0.000 description 1
- 229940112129 campath Drugs 0.000 description 1
- 230000036952 cancer formation Effects 0.000 description 1
- 101150058049 car gene Proteins 0.000 description 1
- 231100000504 carcinogenesis Toxicity 0.000 description 1
- 239000006143 cell culture medium Substances 0.000 description 1
- 230000003915 cell function Effects 0.000 description 1
- 230000010261 cell growth Effects 0.000 description 1
- 239000002771 cell marker Substances 0.000 description 1
- 230000003833 cell viability Effects 0.000 description 1
- 230000005754 cellular signaling Effects 0.000 description 1
- 208000018805 childhood acute lymphoblastic leukemia Diseases 0.000 description 1
- 201000000322 choriocarcinoma of ovary Diseases 0.000 description 1
- 230000001684 chronic effect Effects 0.000 description 1
- 208000025302 chronic primary adrenal insufficiency Diseases 0.000 description 1
- 230000004087 circulation Effects 0.000 description 1
- 238000012411 cloning technique Methods 0.000 description 1
- 239000013599 cloning vector Substances 0.000 description 1
- 238000011260 co-administration Methods 0.000 description 1
- 238000010276 construction Methods 0.000 description 1
- 238000007796 conventional method Methods 0.000 description 1
- 230000002596 correlated effect Effects 0.000 description 1
- 238000009109 curative therapy Methods 0.000 description 1
- 125000000151 cysteine group Chemical group N[C@@H](CS)C(=O)* 0.000 description 1
- 230000002559 cytogenic effect Effects 0.000 description 1
- 102000003675 cytokine receptors Human genes 0.000 description 1
- 108010057085 cytokine receptors Proteins 0.000 description 1
- 206010052015 cytokine release syndrome Diseases 0.000 description 1
- 230000001461 cytolytic effect Effects 0.000 description 1
- 239000002254 cytotoxic agent Substances 0.000 description 1
- 231100000599 cytotoxic agent Toxicity 0.000 description 1
- 230000003013 cytotoxicity Effects 0.000 description 1
- 231100000135 cytotoxicity Toxicity 0.000 description 1
- 108010025198 decaglycine Proteins 0.000 description 1
- 201000002293 dendritic cell sarcoma Diseases 0.000 description 1
- 230000001627 detrimental effect Effects 0.000 description 1
- 238000006471 dimerization reaction Methods 0.000 description 1
- 230000005750 disease progression Effects 0.000 description 1
- 208000035475 disorder Diseases 0.000 description 1
- 230000002222 downregulating effect Effects 0.000 description 1
- 229940079593 drug Drugs 0.000 description 1
- 238000002651 drug therapy Methods 0.000 description 1
- 238000001962 electrophoresis Methods 0.000 description 1
- 208000001991 endodermal sinus tumor Diseases 0.000 description 1
- 210000002472 endoplasmic reticulum Anatomy 0.000 description 1
- 238000005516 engineering process Methods 0.000 description 1
- 108700021358 erbB-1 Genes Proteins 0.000 description 1
- 210000003743 erythrocyte Anatomy 0.000 description 1
- 238000000799 fluorescence microscopy Methods 0.000 description 1
- 230000003325 follicular Effects 0.000 description 1
- 108010063718 gamma-glutamylaspartic acid Proteins 0.000 description 1
- 238000001476 gene delivery Methods 0.000 description 1
- 208000005017 glioblastoma Diseases 0.000 description 1
- 239000003862 glucocorticoid Substances 0.000 description 1
- 108010057083 glutamyl-aspartyl-leucine Proteins 0.000 description 1
- 108010049041 glutamylalanine Proteins 0.000 description 1
- 108010090037 glycyl-alanyl-isoleucine Proteins 0.000 description 1
- 108010019832 glycyl-asparaginyl-glycine Proteins 0.000 description 1
- 108010084264 glycyl-glycyl-cysteine Proteins 0.000 description 1
- 108010079413 glycyl-prolyl-glutamic acid Proteins 0.000 description 1
- 108010020688 glycylhistidine Proteins 0.000 description 1
- 108010077515 glycylproline Proteins 0.000 description 1
- 108010037850 glycylvaline Proteins 0.000 description 1
- 230000002710 gonadal effect Effects 0.000 description 1
- 239000003102 growth factor Substances 0.000 description 1
- 210000002768 hair cell Anatomy 0.000 description 1
- 230000035876 healing Effects 0.000 description 1
- 201000005787 hematologic cancer Diseases 0.000 description 1
- 230000002489 hematologic effect Effects 0.000 description 1
- 208000024200 hematopoietic and lymphoid system neoplasm Diseases 0.000 description 1
- 230000023597 hemostasis Effects 0.000 description 1
- 230000002440 hepatic effect Effects 0.000 description 1
- 208000006359 hepatoblastoma Diseases 0.000 description 1
- 206010073071 hepatocellular carcinoma Diseases 0.000 description 1
- 231100000844 hepatocellular carcinoma Toxicity 0.000 description 1
- 239000000833 heterodimer Substances 0.000 description 1
- 108010040030 histidinoalanine Proteins 0.000 description 1
- 108010018006 histidylserine Proteins 0.000 description 1
- 201000000284 histiocytoma Diseases 0.000 description 1
- 230000006801 homologous recombination Effects 0.000 description 1
- 238000002744 homologous recombination Methods 0.000 description 1
- 108091008039 hormone receptors Proteins 0.000 description 1
- 102000055277 human IL2 Human genes 0.000 description 1
- 206010020718 hyperplasia Diseases 0.000 description 1
- 230000002519 immonomodulatory effect Effects 0.000 description 1
- 230000005965 immune activity Effects 0.000 description 1
- 239000012642 immune effector Substances 0.000 description 1
- 210000000987 immune system Anatomy 0.000 description 1
- 208000026278 immune system disease Diseases 0.000 description 1
- 229940124622 immune-modulator drug Drugs 0.000 description 1
- 208000037495 immunodeficiency 79 Diseases 0.000 description 1
- 230000002163 immunogen Effects 0.000 description 1
- 229940072221 immunoglobulins Drugs 0.000 description 1
- 229940121354 immunomodulator Drugs 0.000 description 1
- 230000004957 immunoregulator effect Effects 0.000 description 1
- 230000000415 inactivating effect Effects 0.000 description 1
- 239000000411 inducer Substances 0.000 description 1
- 208000027866 inflammatory disease Diseases 0.000 description 1
- 238000011221 initial treatment Methods 0.000 description 1
- 230000010354 integration Effects 0.000 description 1
- 102000008616 interleukin-15 receptor activity proteins Human genes 0.000 description 1
- 108040002039 interleukin-15 receptor activity proteins Proteins 0.000 description 1
- 230000009878 intermolecular interaction Effects 0.000 description 1
- 230000008863 intramolecular interaction Effects 0.000 description 1
- 238000010253 intravenous injection Methods 0.000 description 1
- 150000002500 ions Chemical class 0.000 description 1
- 238000002955 isolation Methods 0.000 description 1
- 108010044374 isoleucyl-tyrosine Proteins 0.000 description 1
- 108010027338 isoleucylcysteine Proteins 0.000 description 1
- 238000002372 labelling Methods 0.000 description 1
- 230000003902 lesion Effects 0.000 description 1
- 108010012058 leucyltyrosine Proteins 0.000 description 1
- 201000002364 leukopenia Diseases 0.000 description 1
- 231100001022 leukopenia Toxicity 0.000 description 1
- 206010024627 liposarcoma Diseases 0.000 description 1
- 239000007788 liquid Substances 0.000 description 1
- 210000002809 long lived plasma cell Anatomy 0.000 description 1
- 201000011649 lymphoblastic lymphoma Diseases 0.000 description 1
- 230000000527 lymphocytic effect Effects 0.000 description 1
- 208000003747 lymphoid leukemia Diseases 0.000 description 1
- 108010017391 lysylvaline Proteins 0.000 description 1
- 210000004962 mammalian cell Anatomy 0.000 description 1
- 208000008585 mastocytosis Diseases 0.000 description 1
- 238000005259 measurement Methods 0.000 description 1
- 230000009401 metastasis Effects 0.000 description 1
- 230000001394 metastastic effect Effects 0.000 description 1
- 206010061289 metastatic neoplasm Diseases 0.000 description 1
- 230000000116 mitigating effect Effects 0.000 description 1
- 238000010369 molecular cloning Methods 0.000 description 1
- 239000000178 monomer Substances 0.000 description 1
- 210000005087 mononuclear cell Anatomy 0.000 description 1
- 230000001400 myeloablative effect Effects 0.000 description 1
- 201000005987 myeloid sarcoma Diseases 0.000 description 1
- 230000003039 myelosuppressive effect Effects 0.000 description 1
- 239000013642 negative control Substances 0.000 description 1
- 229910052757 nitrogen Inorganic materials 0.000 description 1
- 238000010899 nucleation Methods 0.000 description 1
- 239000002777 nucleoside Substances 0.000 description 1
- 150000003833 nucleoside derivatives Chemical class 0.000 description 1
- 210000000056 organ Anatomy 0.000 description 1
- 210000003463 organelle Anatomy 0.000 description 1
- 230000002611 ovarian Effects 0.000 description 1
- 201000003707 ovarian clear cell carcinoma Diseases 0.000 description 1
- 239000000137 peptide hydrolase inhibitor Substances 0.000 description 1
- 102000014187 peptide receptors Human genes 0.000 description 1
- 108010011903 peptide receptors Proteins 0.000 description 1
- COLNVLDHVKWLRT-UHFFFAOYSA-N phenylalanine Natural products OC(=O)C(N)CC1=CC=CC=C1 COLNVLDHVKWLRT-UHFFFAOYSA-N 0.000 description 1
- 102000020233 phosphotransferase Human genes 0.000 description 1
- 230000001766 physiological effect Effects 0.000 description 1
- 201000008824 placental choriocarcinoma Diseases 0.000 description 1
- 229920000656 polylysine Polymers 0.000 description 1
- 229920000642 polymer Polymers 0.000 description 1
- 208000005987 polymyositis Diseases 0.000 description 1
- 235000020004 porter Nutrition 0.000 description 1
- 239000013641 positive control Substances 0.000 description 1
- 208000017805 post-transplant lymphoproliferative disease Diseases 0.000 description 1
- 238000012910 preclinical development Methods 0.000 description 1
- 239000002243 precursor Substances 0.000 description 1
- 238000002203 pretreatment Methods 0.000 description 1
- 239000000186 progesterone Substances 0.000 description 1
- 229960003387 progesterone Drugs 0.000 description 1
- 108010077112 prolyl-proline Proteins 0.000 description 1
- 108010093296 prolyl-prolyl-alanine Proteins 0.000 description 1
- 108010031719 prolyl-serine Proteins 0.000 description 1
- 108010079317 prolyl-tyrosine Proteins 0.000 description 1
- 108010004914 prolylarginine Proteins 0.000 description 1
- 108010029020 prolylglycine Proteins 0.000 description 1
- 230000017854 proteolysis Effects 0.000 description 1
- 239000000700 radioactive tracer Substances 0.000 description 1
- 230000010076 replication Effects 0.000 description 1
- 108091008146 restriction endonucleases Proteins 0.000 description 1
- 201000009410 rhabdomyosarcoma Diseases 0.000 description 1
- 208000011581 secondary neoplasm Diseases 0.000 description 1
- 230000035945 sensitivity Effects 0.000 description 1
- 238000012163 sequencing technique Methods 0.000 description 1
- 108010071207 serylmethionine Proteins 0.000 description 1
- 208000000587 small cell lung carcinoma Diseases 0.000 description 1
- MFBOGIVSZKQAPD-UHFFFAOYSA-M sodium butyrate Chemical compound [Na+].CCCC([O-])=O MFBOGIVSZKQAPD-UHFFFAOYSA-M 0.000 description 1
- 229940054269 sodium pyruvate Drugs 0.000 description 1
- 230000002269 spontaneous effect Effects 0.000 description 1
- 206010041823 squamous cell carcinoma Diseases 0.000 description 1
- 238000011255 standard chemotherapy Methods 0.000 description 1
- 238000010254 subcutaneous injection Methods 0.000 description 1
- 239000007929 subcutaneous injection Substances 0.000 description 1
- 238000006467 substitution reaction Methods 0.000 description 1
- 239000000758 substrate Substances 0.000 description 1
- 230000001629 suppression Effects 0.000 description 1
- 208000024891 symptom Diseases 0.000 description 1
- 230000002195 synergetic effect Effects 0.000 description 1
- 230000002381 testicular Effects 0.000 description 1
- 231100001274 therapeutic index Toxicity 0.000 description 1
- 108010033670 threonyl-aspartyl-tyrosine Proteins 0.000 description 1
- 108010072986 threonyl-seryl-lysine Proteins 0.000 description 1
- 230000009258 tissue cross reactivity Effects 0.000 description 1
- 238000012546 transfer Methods 0.000 description 1
- 102000035160 transmembrane proteins Human genes 0.000 description 1
- 108091005703 transmembrane proteins Proteins 0.000 description 1
- 102000027257 transmembrane receptors Human genes 0.000 description 1
- 108091008578 transmembrane receptors Proteins 0.000 description 1
- 102000003298 tumor necrosis factor receptor Human genes 0.000 description 1
- 210000003171 tumor-infiltrating lymphocyte Anatomy 0.000 description 1
- OUYCCCASQSFEME-UHFFFAOYSA-N tyrosine Natural products OC(=O)C(N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-UHFFFAOYSA-N 0.000 description 1
- 108010035534 tyrosyl-leucyl-alanine Proteins 0.000 description 1
- 108010020532 tyrosyl-proline Proteins 0.000 description 1
- 210000003954 umbilical cord Anatomy 0.000 description 1
- 208000022810 undifferentiated (embryonal) sarcoma Diseases 0.000 description 1
- 241001529453 unidentified herpesvirus Species 0.000 description 1
- 238000011144 upstream manufacturing Methods 0.000 description 1
- 210000003462 vein Anatomy 0.000 description 1
- 210000005253 yeast cell Anatomy 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N5/00—Undifferentiated human, animal or plant cells, e.g. cell lines; Tissues; Cultivation or maintenance thereof; Culture media therefor
- C12N5/06—Animal cells or tissues; Human cells or tissues
- C12N5/0602—Vertebrate cells
- C12N5/0634—Cells from the blood or the immune system
- C12N5/0636—T lymphocytes
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/46—Cellular immunotherapy
- A61K39/461—Cellular immunotherapy characterised by the cell type used
- A61K39/4611—T-cells, e.g. tumor infiltrating lymphocytes [TIL], lymphokine-activated killer cells [LAK] or regulatory T cells [Treg]
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/46—Cellular immunotherapy
- A61K39/461—Cellular immunotherapy characterised by the cell type used
- A61K39/4613—Natural-killer cells [NK or NK-T]
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/46—Cellular immunotherapy
- A61K39/463—Cellular immunotherapy characterised by recombinant expression
- A61K39/4631—Chimeric Antigen Receptors [CAR]
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/46—Cellular immunotherapy
- A61K39/464—Cellular immunotherapy characterised by the antigen targeted or presented
- A61K39/4643—Vertebrate antigens
- A61K39/4644—Cancer antigens
- A61K39/464402—Receptors, cell surface antigens or cell surface determinants
- A61K39/464411—Immunoglobulin superfamily
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/46—Cellular immunotherapy
- A61K39/464—Cellular immunotherapy characterised by the antigen targeted or presented
- A61K39/4643—Vertebrate antigens
- A61K39/4644—Cancer antigens
- A61K39/464402—Receptors, cell surface antigens or cell surface determinants
- A61K39/464416—Receptors for cytokines
- A61K39/464417—Receptors for tumor necrosis factors [TNF], e.g. lymphotoxin receptor [LTR], CD30
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/46—Cellular immunotherapy
- A61K39/464—Cellular immunotherapy characterised by the antigen targeted or presented
- A61K39/4643—Vertebrate antigens
- A61K39/4644—Cancer antigens
- A61K39/464402—Receptors, cell surface antigens or cell surface determinants
- A61K39/464416—Receptors for cytokines
- A61K39/464419—Receptors for interleukins [IL]
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/46—Cellular immunotherapy
- A61K39/464—Cellular immunotherapy characterised by the antigen targeted or presented
- A61K39/4643—Vertebrate antigens
- A61K39/4644—Cancer antigens
- A61K39/464402—Receptors, cell surface antigens or cell surface determinants
- A61K39/464429—Molecules with a "CD" designation not provided for elsewhere
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
- A61P35/02—Antineoplastic agents specific for leukemia
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P43/00—Drugs for specific purposes, not provided for in groups A61P1/00-A61P41/00
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/52—Cytokines; Lymphokines; Interferons
- C07K14/54—Interleukins [IL]
- C07K14/5443—IL-15
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/705—Receptors; Cell surface antigens; Cell surface determinants
- C07K14/70503—Immunoglobulin superfamily
- C07K14/7051—T-cell receptor (TcR)-CD3 complex
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/705—Receptors; Cell surface antigens; Cell surface determinants
- C07K14/70503—Immunoglobulin superfamily
- C07K14/70514—CD4
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/705—Receptors; Cell surface antigens; Cell surface determinants
- C07K14/70503—Immunoglobulin superfamily
- C07K14/70517—CD8
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/705—Receptors; Cell surface antigens; Cell surface determinants
- C07K14/70503—Immunoglobulin superfamily
- C07K14/70521—CD28, CD152
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/705—Receptors; Cell surface antigens; Cell surface determinants
- C07K14/70578—NGF-receptor/TNF-receptor superfamily, e.g. CD27, CD30, CD40, CD95
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/2803—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against the immunoglobulin superfamily
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/2866—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against receptors for cytokines, lymphokines, interferons
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/2878—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against the NGF-receptor/TNF-receptor superfamily, e.g. CD27, CD30, CD40, CD95
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/2887—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against CD20
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/289—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against CD45
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/2896—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against molecules with a "CD"-designation, not provided for elsewhere
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N5/00—Undifferentiated human, animal or plant cells, e.g. cell lines; Tissues; Cultivation or maintenance thereof; Culture media therefor
- C12N5/06—Animal cells or tissues; Human cells or tissues
- C12N5/0602—Vertebrate cells
- C12N5/0634—Cells from the blood or the immune system
- C12N5/0646—Natural killers cells [NK], NKT cells
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K2239/00—Indexing codes associated with cellular immunotherapy of group A61K39/46
- A61K2239/31—Indexing codes associated with cellular immunotherapy of group A61K39/46 characterized by the route of administration
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K2239/00—Indexing codes associated with cellular immunotherapy of group A61K39/46
- A61K2239/38—Indexing codes associated with cellular immunotherapy of group A61K39/46 characterised by the dose, timing or administration schedule
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K2239/00—Indexing codes associated with cellular immunotherapy of group A61K39/46
- A61K2239/46—Indexing codes associated with cellular immunotherapy of group A61K39/46 characterised by the cancer treated
- A61K2239/48—Blood cells, e.g. leukemia or lymphoma
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/01—Fusion polypeptide containing a localisation/targetting motif
- C07K2319/02—Fusion polypeptide containing a localisation/targetting motif containing a signal sequence
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/01—Fusion polypeptide containing a localisation/targetting motif
- C07K2319/03—Fusion polypeptide containing a localisation/targetting motif containing a transmembrane segment
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/50—Fusion polypeptide containing protease site
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/95—Fusion polypeptide containing a motif/fusion for degradation (ubiquitin fusions, PEST sequence)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2510/00—Genetically modified cells
Abstract
본 발명은 키메라 항원 수용체 (CAR) 폴리펩타이드에 관련되는 조성물 및 방법 및 이에 연관되는 방법에 관한 것이다. 한 실시 양태에서, 본 발명은 적어도 2개의 표적에 지시되는 키메라 항원 수용체 폴리펩타이드를 갖는 조작된 세포에 관한 것이다. 또 다른 실시 양태에서, 본 발명은 키메라 항원 수용체 폴리펩타이드 및 인핸서 모이어티를 갖는 조작된 세포에 관한 것이다.
Description
<관련 출원에 대한 교차-참조>
본 출원은 미국 가출원 제62/184,321호 (2015년 6월 25일자 출원), 62/235,840호 (2015년 10월 1일자 출원), 및 62/244, 435호 (2015년 10월 21일자 출원)으로부터 우선권을 주장하는 국제 PCT 출원이며, 이들 모두는 그 전문이 본원에 참조로 포함된다.
림프구의 일종인 T 세포는 세포 매개 면역에 중심적인 역할을 한다. 그들은 세포 표면의 T 세포 수용체 (TCR)의 존재로 B 세포 및 자연 살상(natural killer) 세포 (NK 세포)와 같은 다른 림프구와 구별된다. CD4 + T 또는 CD4 T 세포라고도 불리는 T 헬퍼 세포는 그의 표면에 CD4 당단백질을 발현한다. 헬퍼 T 세포는 MHC (주조직 적합 복합체) 클래스 II 분자에 의해 제시되는 펩타이드 항원에 노출될 때 활성화된다. 일단 활성화되면, 이들 세포는 급속하게 증식하고 면역 반응을 조절하는 사이토카인을 분비한다. CD8 + T 세포 또는 CD8 T 세포로도 알려진 세포독성 T 세포는 세포 표면에 CD8 당단백질을 발현한다. CD8 + T 세포는 MHC 클래스 I 분자에 의해 제시되는 펩티드 항원에 노출될 때 활성화된다. T 세포의 하위세트인 기억 T 세포는 장기간 지속되다가 그들의 동족 항원에 반응하므로 면역 시스템에 과거의 감염 및/또는 종양 세포에 대한 "기억"을 제공한다.
T 세포는 그의 표면 상에 키메라 항원 수용체 (CAR)라고 불리는 특정 수용체를 생산하도록 유전적으로 조작될 수 있다. CAR은 T 세포가 종양 세포의 특정 단백질 (항원)을 인식할 수 있게 해주는 단백질이다. 이러한 조작된 CAR T 세포는 그 다음에 세포 수자가 수십억에 달할 때까지 실험실에서 배양된다. 팽창된 CAR T세포 집단은 그 다음에 환자에게 주입된다.
지금까지의 임상 시험들은 키메라 항원 수용체 (CAR) T 세포가 표준화학요법에 내성을 보이는 혈액 악성종양에 있어서 큰 가능성을 가진다는 점을 보여 주었다. 특히, CD19에 특이적인 CAR(CD19CAR) T 세포 요법이 B세포 악성종양을 장기적으로 완화시키는 것을 비롯한 놀라운 결과를 가져왔다 (Kochenderfer, Wilson et al. 2010, Kalos, Levine et al. 2011, Porter, Levine et al. 2011, Davila, Riviere et al. 2013, Grupp, Frey et al. 2013, Grupp, Kalos et al. 2013, Kalos, Nazimuddin et al. 2013, Kochenderfer, Dudley et al. 2013, Kochenderfer, Dudley et al. 2013, Lee, Shah et al. 2013, Park, Riviere et al. 2013, Maude, Frey et al. 2014).
B세포 백혈병과 림프종에 대한 CAR 요법의 성공에도 불구하고, T 세포 악성종양에 대한 CAR 요법의 적용은 아직 잘 확립되지 않았다. T 세포 악성종양이 B 세포 악성종양과 비교할 때 극적으로 보다 불량한 결과와 연관되는 것을 고려하면 (Abramson, Feldman et al. 2014), CAR 요법은 이 점에서 큰 임상적 수요를 더욱 부각시킬 잠재력을 갖고 있다.
지금까지, 현재 노력은 다양한 B-세포 악성 종양에 대한 CAR T-세포 효능을 입증하는 데 집중하고 있다. CD19CAR을 사용하는 B-ALL에서는 약 90%의 초기 완화율이 일반적이지만 이들 대부분은 1년 이내에 재발한다. 재발은 항원 회피로 인해 적어도 부분적으로 발생한다. 따라서, 재발을 방지하기 위한 더욱 효과적인 CAR T 세포 치료방법이 시급히 필요하다. 효과적인 CAR 디자인을 보장하거나 가이드하는 일반적인 규칙이 없기 때문에 표적 발견 및 선택이 초기 단계이다.
CAR 치료 접근법의 더 광범위한 채택을 저해하는 일부 장애물들이 있다. 가장 일반적인 과제로는 (1) 항원 표적 및 키메라 항원 수용체(들)의 선택; (2) CAR 디자인; (3) 종양 이질성, 특히 종양 항원의 표면 발현에서의 변이이다. 단일 항원을 표적으로 하는 것은 면역 회피의 위험요소를 가지며, 이는 다수의 원하는 항원을 표적으로 함으로써 극복될 수 있다.
대부분의 CAR 키메라 항원 수용체는 단클론항체로부터 유래된 scFvs이며, 이들 단클론항체 중 일부는 임상 시험 또는 질환의 치료에 사용되고 있다. 하지만 이들 효과는 제한적이므로 이는 CAR과 같은 대안적이고 더욱 강력한 표적화 접근이 요구된다. scFvs는 CAR에 대해 가장 통상적으로 사용되는 키메라 항원 수용체이다. 하지만, CAR 친화성 결합 및 항원 상의 인식된 에피토프의 위치는 기능에 영향을 줄 수 있다. 또한, T 세포 또는 NK 세포 상의 표면 CAR 발현 수준은 적절한 리더 서열과 프로모터에 의해 영향을 받는다. 더욱이 과발현된 CAR 단백질은 세포에 유독성을 나타낼 수 있다.
그러므로, T- 세포 연관 악성 종양에 대한 더욱 효과적이고 안전하며 그리고 효율적인 표적화를 가능하게 하는 개선된 키메라 항원 수용체 - 기반 요법에 대한 필요성이 남아있다.
하나의 실시양태에서, 본 개시내용은 제1 항원 인식 도메인, 제1 신호 펩타이드, 제1 힌지 영역, 제1 막통과(transmembrane) 도메인, 제1 보조 자극 도메인 및 제1 신호전달 도메인을 포함하는 제1 키메라 항원 수용체 폴리펩타이드와 제2 항원 인식 도메인, 제2 신호 펩타이드, 제2 힌지 영역, 제2 막통과 도메인, 제2 보조 자극 도메인 및 제2 신호전달 도메인을 포함하는 제2 키메라 항원 수용체 폴리펩타이드를 가지며, 상기 제1 항원 인식 도메인은 제2 항원 인식 도메인과 상이한 것인 조작된 세포를 제공한다.
또 다른 하나의 실시양태에서, 본 개시내용은 키메라 항원 수용체 및 인핸서를 포함하는 조작된 폴리펩타이드를 제공한다.
또 다른 하나의 실시양태에서, 본 개시내용은 키메라 항원 수용체 폴리펩타이드 및 인핸서를 포함하는 조작된 폴리펩타이드를 제공한다.
또 다른 하나의 실시양태에서, 본 개시내용은 신호 펩타이드, CD45 항원 인식 도메인, 힌지 영역, 막통과 도메인, 적어도 하나의 보조 자극 도메인 및 신호전달 도메인을 포함하는 조작된 키메라 항원 수용체 폴리펩타이드를 제공한다. 또 다른 하나의 실시양태에서, 본 개시내용은 전술한 폴리펩타이드를 코딩하는 폴리뉴클레오타이드를 제공한다.
또 다른 하나의 실시양태에서, 본 개시내용은 전술한 조작된 폴리펩타이드 또는 폴리뉴클레오타이드를 가지는 조작된 세포를 제공한다.
또 다른 하나의 실시양태에서, 본 개시내용은 다음 단계를 포함하는 표적 세포의 수를 감소시키는 방법을 제공한다. (i.) 상기 표적 세포를 적어도 하나의 키메라 항원 수용체 폴리펩타이드를 가지는 조작된 세포의 유효량과 접촉시키는 단계 (다중 키메라 항원 수용체 폴리펩타이드를 가지는 조작된 세포에 대해, 각 키메라 항원 수용체 폴리펩타이드는 독립적임); 및 (ii.) 선택적으로, 상기 세포의 수의 감소를 분석하는 단계. 표적 세포는 인터루킨 6 수용체, NY-ESO-1, 알파 태아단백 (AFP), 글리피칸-3 (GPC3), BAFF-R, BCMA, TACI, LeY, CD5, CD13, CD14, CD15, CD19, CD20, CD22, CD33, CD41, CD45, CD61, CD64, CD68, CD117, CD123, CD138, CD267, CD269, CD38, Flt3 수용체 및 CS1로 이루어진 그룹으로부터 선택된 적어도 하나의 세포 표면 항원을 포함한다.
또 다른 하나의 실시 양태에서, 본 개시내용은 상기 기술된 임의의 조작된 세포를 이를 필요로 하는 환자에 투여함으로써 B-세포 림프종, T-세포 림프종, 다발성 골수종, 만성 골수성 백혈병, B-세포 급성 림프모구성 백혈병 (B-ALL) 및 세포증식성 질환을 치료하는 방법을 제공한다.
도 1. cCAR 구축물의 도식적 표현 (이하 "다중 CAR 또는 복합 CAR"). 다중 또는 복합 CAR은 다중 항원 (예: 세포 유형1 또는 세포 유형2 또는 동일한 세포 유형)을 표적으로 한다. 다중 또는 cCAR T 세포 면역 요법은 상이한 또는 동일한 항원 인식 도메인, 힌지 영역, 막통과 도메인, 다양한 보조 자극 도메인(들) 및 세포 내 신호 전달 도메인을 포함하는 개개의 구성 요소 CAR을 포함한다.
도 2A. cCAR-T 구축물의 도식적 표현. 상기 구축물은 P2A 펩타이드에 의해 연결된 CAR의 다중 모듈 단위의 발현을 유도하는 SFFV 프로모터를 포함한다. 링커가 절단되면, cCAR들은 분리되고 CD33 및/또는 CD123을 발현하는 표적에 결합한다. 새로운 cCAR 구축물로서, 이 구축물의 활성화 도메인은 CD33 CAR 단편 상의 4-1BB 및 CD123 CAR 상의 CD28 영역을 포함할 수 있으나, 이에 한정되는 것은 아니다.
도 2B. 형질도입된 CD33CD123 cCAR-T 세포의 발현을 도시하는 웨스턴 블롯. 이 도면은 2 개의 상이한 CAR 단백질, 즉, CD33 CAR 및 CD123 CAR의 발현을 도시한다. 링커가 절단되면 CD33 및 CD123 CAR 모두를 발현하는 cCAR-T 세포는 두 개의 명확하고 일관되게 색이 매우 짙은 단백질 밴드를 생성한다. 녹색 형광 단백질 (GFP)은 음성 대조군으로서 포함된다.
도 2C. 형질도입의 효율을 나타내는 유동 세포측정법. 상단 패널은 UCB (제대혈) 및 PB (말초 혈액) T 세포에 사용하기 전 최대 형질 도입 효율을 측정하기 위해 293FT HEK (인간 배아 신장) 세포 상에서 시험된 CD33CD123 cCAR (CD33CD123-2G-CAR라고도 지칭됨)에 대한 렌티바이러스의 역가를 보여준다. 하단 패널은 CD33CD123 cCAR 구축물을 포함하는 렌티바이러스 벡터로 형질 도입된 CD33CD123 cCAR (CD33CD123-2G-CAR 라고도 지칭됨) T- 세포, 및 대조군으로서 GFP- 형질 도입된 세포를 보여준다. 노란색 원에 의해 표시되는 백분율은 형질 도입 효율에 대한 프록시이다.
도 3. 고효율 화합물 CAR (cCAR) 생성 방법을 보여주는 도식.
도 4. CD33CD123-2G CAR-T 세포 (cCAR)의 전골수구성 백혈병 세포주 HL60와의 인큐베이션을 나타내는 공동 배양 분석. cCAR-T 세포 (하부 패널)는 대조 GFP 형질 도입된 T 세포 (상부 패널)와 비교된다. 살상의 효능은 약 24 시간 동안 배양한 후에 남겨진 CD33+ 세포의 집단에 의해 측정된다 (노란색 원으로 둘러싸임).
도 5. cCAR-T 세포의 약 100% CD33과 약 50-80% CD123을 발현하는 골수성 백혈병 세포주 KG-1a와의 인큐베이션을 나타내는 공동 배양 분석. cCAR-T 세포 (하부 패널)는 대조 GFP 형질 도입된 T 세포 (상부 패널)와 비교된다. 살상의 효능은 약 24시간 동안 배양한 후에 남겨진 CD33+ 세포의 집단에 의해 측정된다.
도 6. cCAR-T 세포의 AML 환자 샘플 (여기서는 AML-9라고 지칭됨)과의 인큐베이션을 나타내는 공동 배양 분석. 환자 세포는 예를 들어 백혈병 세포, 단핵구 및 다른 유형의 모세포와 같은 혼합된 세포 집단을 포함한다. CD33은 백혈병 세포에 대한 특이적 마커인 CD34와 마찬가지로 CAR-T 작용에 대한 마커로서 또한 작용한다. CAR-T 패널 (우측)은 대조 GFP 형질 도입된 T 세포 (중간)와 비교된다. 살상의 효능은 적어도 24시간 동안 인큐베이션한 후에 남겨진 CD33+/CD34+ 세포의 집단에 의해 측정된다.
도 7. cCAR-T 세포의 B-ALL 환자 샘플 (여기서는 Sp-BM-B6이라고 지칭됨)과의 인큐베이션을 나타내는 공동 배양 분석. 환자 세포는 예를 들어, 백혈병 세포, 단핵구 및 다른 유형의 모세포와 같은 혼합된 세포 집단을 포함한다. CD34는 백혈병 세포에 대한 특이적 마커로서 작용한다. CAR-T 패널 (우측)은 대조 GFP 형질 도입된 T 세포 (중간)와 비교된다. 살상의 효능은 적어도 24시간 동안 인큐베이션한 후에 남겨진 CD34+ 세포 집단에 의해 측정된다.
도 8. NK-92 세포에서의 CD33CD123 cCAR 발현. CD33CD123 cCAR 발현은 염소-항-마우스 항체, F(ab)2를 사용하여 검출된다.
도 9. cCAR NK-92 세포의 HL-60과의 인큐베이션을 나타내는 공동 배양 분석. cCAR NK-92 세포는 GFP 형질 도입된 NK-92 세포와 비교된다. 살상의 효능은 약 24시간 동안 인큐베이션한 후 남은 CD33+ 세포의 집단에 의해 측정된다.
도 10. cCAR NK-92 세포의 KG1a와의 인큐베이션을 나타내는 공동 배양 분석. cCAR NK 세포 패널은 GFP 형질 도입된 NK-92 세포와 비교된다. 살상의 효능은 약 24시간 동안 인큐베이션한 후 남은 CD33+ 세포의 집단에 의해 측정된다.
도 11. HL-60 또는 KG1a을 사용한 CD33CD123 cCAR (CAR-CD33/123) NK-92 세포의 용량 반응.
살상의 효능은 약 24시간 동안 인큐베이션한 후 남은 CD33+ 세포의 집단에 의해 측정된다.
도 12. KG11 세포의 두 집단에서의 CD33CD123 cCAR NK-92 세포의 살상 능력과 대조의 비교. CAR-CD33/123 (CD33CD123 cCAR NK-92 세포) 와 표적 세포 kG1a의 상이한 비율로 분석을 수행하였다. 살상의 효능은 약 24시간 동안 인큐베이션한 후 남은 CD33+CD123+ 또는 CD33+CD123- 세포의 집단에 의해 측정된다.
도 13. cCAR의 도식적 표현. 구축물은 링커에 의해 연결된 CAR의 다중 모듈 단위의 발현을 유도하는 SFFV 프로모터를 포함한다. 링커가 절단되면, cCAR들은 분리되고 CD19 및/또는 CD20, 및/또는 CD22 및/또는 138의 다양한 표적 항원의 조합을 발현하는 표적에 결합한다. 다중 cCAR은, 예를 들어 4-1BB (4-BB로도 표기함) 및/또는 CD28로 제한하는 것 없이, 동일하거나 또는 상이한 보조 자극 도메인을 이용한다.
도 14 A-C. BCMA-CS1 cCAR 구축물 도식 (BC1cCAR). (A) 구축물은 P2A 펩타이드에 의해 연결된 CAR의 2 개의 모듈 단위의 발현을 유도하는 SFFV 프로모터로 구성된다. 이 P2A 펩티드가 절단되면, cCAR들은 분리되고 BCMA 및/또는 CS1을 발현하는 표적에 결합한다. 2개의 CAR 단위는 동일한 보조 자극 도메인 4-1BB를 사용한다. (B) 벡터 (왼쪽)와 BC1cCAR (오른쪽, 사각형으로 강조됨)에 대한 T 세포 표면에서의 BC1cCAR 발현의 유동 세포측정법 분석이며, BC1cCAR은 F(Ab)2에 대해 15.3 %의 양성을 보임. 동형 대조에 대해 게이팅함. (C) BCMA cDNA (BCMA-K562) (NIH, Kochenderfer로부터 얻음)로 형질 도입한 K562 세포와의 공동 배양으로 BC1cCAR-T 세포의 예비 기능 검증. 막대 그래프는 대조 T 세포에 대한 BCMA-K562 세포주의 용해 그리고 대조에 대한 야생형 K562 (wt-K562)의 용해를 보여준다.
도 14D. 각 단위에 대해 4-1BB 또는 CD28의 2개의 상이한 보조 자극 도메인을 사용하는 BCMA-CS1-2G 구축물. 이 구축물은 P2A 펩타이드에 의해 연결된 CAR의 2개의 모듈 단위의 발현을 유도하는 SFFV 프로모터를 포함한다. 이 P2A 펩타이드가 절단되면, cCAR들은 분리되고 BCMA 및/또는 CS1을 발현하는 표적에 결합한다. 2개의 CAR 단위는 4-1BB 또는 CD28의 서로 다른 보조 자극 도메인을 사용한다. 벡터 (왼쪽)와 BC1cCAR (오른쪽, 사각형으로 강조됨)에 대한 T 세포 표면에서의 BC1cCAR 발현의 유동 세포측정법 분석이며, BC1cCAR은 F(Ab)2에 대해 희귀한 양성 세포를 보임. 동형 대조에 대해 게이팅함.
도 14E. HEK-293FT 세포에서의 BC1cCAR 및 BCMA-CS1-2G의 단백질 발현. HEK-293FT 세포를 GFP (레인 1), BC1cCAR (레인 2), CD269-CS1-2G (레인 3)를 위한 렌티바이러스 플라스미드로 형질 감염시키고, 형질 감염 48시간 후에 상청액을 제거하고 세포를 또한 분리하였다. 웨스턴 블롯을 위하여 세포를 용해하고 마우스 항-인간 CD3z 항체로 프로브하였다.
도 15A-B. MM1S 세포주 공동배양. 공동배양을 24시간 이내에 수행하고 수집하고 유동 세포측정법을 통해 분석하였다. 표적 MM1S 세포 (골수종 세포)를 Cytotracker (CMTMR) 염료로 표지하여 효과 T 세포로부터 구별하였다. 항 BCMA (CD269) 및 항-CS1 (CD319) 항체로 집단을 게이팅하였다. 도 15A: 공동 배양의 유동 세포측정법 도시. 도 15B: 오른쪽: 용해 대 E:T 비율의 그래픽 요약.
도 16A-B. RPMI-8226 세포주 공동 배양. 공동 배양을 24시간 이내에 수행하고 수집하고 유동 세포측정법을 통해 분석하였다. 표적 RPMI-8226 세포를 Cytotracker (CMTMR) 염료로 표지하여 효과 T 세포로부터 구별하였다. 항-BCMA (CD269) 및 항-CS1 (CD319) 항체로 집단을 게이팅하였다. 도 16A: 공동 배양의 유동 세포측정법 묘사. 도 16B: 용해 대 E:T 비율의 그래픽 요약.
도17 A-B. U266 세포주 공동 배양. 공동 배양을 24 시간 이내에 수행하고 수집하고 유동 세포측정법을 통해 분석하였다. 표적 U266 세포를 Cytotracker (CMTMR) 염료로 표지하여 효과 T 세포로부터 구별하였다. 항 BCMA (CD269) 및 항-CS1 (CD319) 항체로 집단을 게이팅하였다. (A) 공동 배양의 유동 세포측정법 도시. (B) 용해 대 E:T 비율의 그래픽 요약.
도 18A-B. MM10-G 원발성 환자 샘플 공동 배양 및 특이적 용해. 공동 배양을 24시간 이내에 수행하고 수집하고 유동세포측정법을 통해 분석하였다. 표적 MM10-G 세포를 Cytotracker (CMTMR) 염료로 표지하여 효과 T 세포로부터 구별하였다. 항 BCMA (CD269) 및 항-CS1 (CD319) 항체로 집단을 게이팅하였다. 특히, 게이팅은 MM10-G가 뚜렷한 BCMA+ 및 CS1+ 집단을 나타냄을 보여준다. 도 18A: 공동 배양의 유동 세포측정법 도시. 도 18B: 용해 대 E:T 비율의 그래픽 요약.
도 19A-B. MM7-G 원발성 환자 샘플 공동 배양 및 특이적 용해. 공동 배양을 24시간 이내에 수행하고 수집하고 유동 세포측정법을 통해 분석하였다. 표적 MM7-G 세포를 Cytotracker (CMTMR) 염료로 표지하여 효과 T 세포로부터 구별하였다. 항-BCMA (CD269) 및 항-CS1 (CD319) 항체로 집단을 게이팅하였다. 도 19A: 공동 배양의 유동 세포측정법 도시. 도 19B: 용해 대 E:T 비율의 그래픽 요약.
도 20A-B. MM11-G 원발성 환자 샘플 공동 배양 및 특이적 용해. 공동 배양을 24 시간 이내에 수행하고 수집하고 유동 세포측정법을 통해 분석하였다. 표적 MM11-G 세포를 Cytotracker (CMTMR) 염료로 표지하여 효과 T 세포로부터 구별하였다. 항-BCMA (CD269) 및 항-CS1 (CD319) 항체로 집단을 게이팅하였다. 도 20A: 공동 배양의 유동세포측정법 도시. 도 20B: 용해 대 E:T 비율의 그래픽 요약.
도 21. CD269-CS1-BBCAR NK 세포는 생체내에서 항-백혈병 효과를 나타낸다. NSG 마우스에게 준치사적 방사선을 조사하고 다음 날 루시페레이스를 발현하는 MM.1S 다발성 골수종 세포를 정맥내 주사하여 측정 가능한 종양 형성을 유도하였다. 3 일 후, 마우스에게 8 x 106 CD269-CS1-BBCAR NK 세포 또는 벡터 대조 NK 대조 세포를 정맥내 주사하였다. 제 3, 6, 및 8 일에 마우스에게 RediJect D-Luciferin을 피하 주사하여 IVIS 영상을 시행하였다. CD269-CS1-BBCAR NK 주사된 마우스에 대해 측정된 평균 광 강도를 벡터 대조 NK 주사된 마우스의 것과 비교하였다.
도 22. 도 21의 연구에 근거하여 마우스의 생존율을 측정하고 두 그룹 사이에서의 마우스의 생존율을 비교하였다.
도 23. CRISPR / Cas9 간섭 시스템. sgRNA 및 Cas9 퓨로마이신의 발현은 U6 및 SFFV 프로모터에 의해 각각 유도된다. Cas9는 E2A 자가절단 서열에 의해 퓨로마이신 내성유전자와 연결되어 있다.
도 24. 혈액 악성 종양을 표적으로하는 CAR T 또는 NK 세포의 생성을 위한 단계의 예를 제공하는 도식.
도 25. CRISPR/Cas9 렌티바이러스 시스템을 사용한 안정된 CD45 녹다운 NK-92 세포의 생성 및 세포 분류. 유동 세포측정법 분석은 NK-92 세포 표면 상에서의 CD45 발현 수준을 나타내었다 (왼쪽 패널). sgCD45B CRISPR을 NK-92 세포에 형질도입한 후, 형질도입된 세포를 퓨로마이신을 함유하는 배지 중에서 수주 동안 배양하였다. CD45 음성 NK-92 세포를 CD45 항체를 사용하여 결정하고 분류하였다. 안정된 NK45i-92 (CD45 녹다운) NK-92 세포의 순도는 유동 세포측정법 분석에 의해 측정되었다 (오른쪽 패널). 이 데이터는 NK45i-92 세포가 성공적으로 생성되고 얻어졌다는 것을 보여 주었다.
도 26. 야생형, GFP 형질도입된 NK-92 또는 NK45i-92NK 세포의 세포 성장 곡선. NK-92 세포에서의 CD45- 녹다운 (KD)에 의해 야기된 세포 증식에 대한 효과를 평가하기 위해, 24 웰 플레이트 (well plates)에 시딩한 후 48 시간 및 96 시간에 NK-92 (●), GFP-형질도입된 NK-92 (■) 및 NK45i-92 (▲)의 세포 수를 계수하였다. 48시간 시점에 IL-2를 첨가하였다 (n=3회의 독립적인 실험을 반복으로 수행하였다). 데이터는 평균치+/- S.D. 이다. 이들 데이터는 NK-92상의 CD45 수용체의 녹다운이 비형질도입된 NK-92 또는 GFP-형질도입된 NK-92 세포와 비교하여 유사한 세포 성장 곡선을 보인다는 것을 나타냈다.
도 27 A-B. CCRF-CEM (표적: T) 및 GFP NK-92 또는 GFP NK45i-92 세포 (효과기: E), 5:1 (E:T) 비율의 공동 배양 분석. 16시간 인큐베이션. (A) CCRF-CEM 단독 (좌측 패널의 파란색 점), CCRF-CEM과 대조 GFP 형질 도입된 NK-92 세포의 공동 배양 (중간 패널) 또는 CCRF-CEM과 GFP NK45i-92 세포의 공동 배양 (우측 패널)의 유동 세포측정법 분석. 모든 패널에서의 파란색 점은 남아있는 표적 CCRF-CEM 세포를 나타내고 빨간색 점은 공동 배양 분석에 의한 효과 세포를 나타낸다. 인큐베이션 시간은 전부 16 시간이었고 효과 T 세포: 표적 세포의 비율은 5:1이었다. 모든 실험은 반복으로 수행되었다. (B) 막대 그래프는 CCRF-CEM과의 공동 배양 분석에서 GFP 형질도입된 NK45i-92 세포에 의한 세포 용해의 백분율을 대조 GFP 형질도입된 NK92 세포와 비교하여 보여준다. 이들 데이터는 시험관내 공동 배양 분석에서 NK-92 세포에서의 CD45의 녹다운이 GFP-대조 NK-92 세포에 비교하여 CCRF-CEM 세포에 대한 살상 활성의 유의한 차이를 나타내지 않는다는 것을 시사한다. 파란색 점은 왼쪽 상단 4분면에 있다.
도 28 A-B. CCRF-CEM (표적: T) 그리고 GFP NK-92, CD5CAR NK-92 또는 CD5CAR NK45i-92 세포 (효과기: E)와의 공동 배양 분석. 5: 1 (E: T) 비율. 16 시간 인큐베이션. (A) 오른쪽에서 왼쪽으로, CCRF-CEM 단독 (왼쪽 패널), CCRF-CEM과 대조 GFP NK-92 세포의 공동 배양 (중간 왼쪽 패널), CCRF-CEM과 CD5CAR NK-92 세포의 공동 배양 (중간 오른쪽 패널), CCRF-CEM과 CD5CAR NK45i-92 세포의 공동 배양 (오른쪽 패널)의 유동 세포측정법 분석. 모든 패널의 파란색 점은 남아있는 표적 CCRF-CEM 세포를 나타내고 빨간색 점은 공동 배양 분석에 의한 효과 세포를 나타낸다. 인큐베이션 시간은 전부 16시간이었고 효과 T 세포: 표적 세포의 비율은 5:1이었다. 모든 실험은 반복으로 수행되었다. (B) 막대 그래프는 CCRF-CEM과의 공동 배양 분석에서 CD5CAR NK-92 세포 또는 CD5CAR NK45i-92 세포에 의한 세포 용해의 백분율을 대조 GFP NK92 세포와 비교하여 보여준다. 데이터는 평균치 +/- S.D. 이다. CD5CAR NK 세포 및 CD5CAR NK45i-92 세포 모두 대조 GFP NK-92 세포와 비교하여 CD5-양성 CCRF-CEM에 대해 100%에 가까운 세포 살상 활성을 나타낸다. 이들 데이터는 CD5CAR NK 세포 및 CD5CAR NK45i-92 세포가 GFP-대조 NK-92 세포와 비교하여 시험관내 공동 배양 분석에서 CD5를 발현하는 CCRF-CEM 세포를 효과적으로 용해시킬 수 있고, CD45의 녹다운이 NK-92 세포에서의 살상 활성을 위한 세포 기능에 영향을 미치지 않는다는 증거를 제공한다. 파란색 점은 왼쪽으로부터 시작하여 처음 두 패널의 왼쪽 상단 4분면에 있다.
도 29A-B. CD45CAR 구축물의 구성과 그 발현. (A) CD45CAR 렌티바이러스 벡터의 도식적 표시. CD45CAR 구축물은 모듈화된 신호전달 도메인이고 리더 서열, 항-CD45scFv, 힌지 도메인 (H), 막통과 도메인 (TM), 구축물의 제3세대 CAR로서의 특징인 두 개의 보조 자극 도메인 (CD28 및 4-1BB), 및 세포내 신호전달 도메인 CD3 제타를 포함한다. (B), HEK-293FT 세포를 GFP (레인 1) 및 CD45CAR (레인 2)를 위한 렌티바이러스 플라스미드로 형질 감염시켰다. 형질 감염 48시간 후에 상청액을 제거하고 세포도 분리하였다. 웨스턴 블롯을 위해 세포를 용해시키고 마우스 항-인간 CD3z 항체로 프로브하였다.
도 30 A-B. NK45i-92 세포로의 CD45CAR의 형질도입 및 CD45CAR 형질도입된 세포의 세포분류. (A) CD45CAR 렌티바이러스가 NK45i-92 세포로 형질도입된 후에 NK45i-92상의 CD45CAR의 발현 수준을 유동 세포측정법 분석 (중간 패널에서 파란 동그라미로 표시)으로 NK45i-92 세포 (왼쪽 패널)와 비교하여 측정하였다. CD45CAR 발현된 NK45i-92 세포를 분류하고 세포 표면 상의 CD45 발현 수준을 유동 세포측정법 분석 (오른쪽 패널)에 의해 측정하였다. (B) 유동 세포측정법 분석에 의해 검출된 세포 표면 상 CD45CAR 발현은 약 87%였다.
도 31A-B. CCRF-CEM (표적: T) 와 GFP NK-92 또는 CD45CAR NK45i-92 세포 (효과기: E)와의 공동 배양 분석. 5:1 (E:T) 비율. 16 시간 인큐베이션. (A) CCRF-CEM과 대조 GFP 형질도입된 NK-92 세포의 공동 배양 (왼쪽 패널) 또는 CCRF-CEM과 CD45CAR NK45i-92 세포의 공동 배양 (오른쪽 패널)의 유동 세포측정법 분석. 모든 패널의 파란색 점은 남아있는 표적 CCRF-CEM 세포를 나타내고 빨간색 점은 공동 배양 분석에 의한 효과기 NK-92 세포를 보여준다. 모든 인큐베이션 시간은 16 시간이었고 효과 T세포: 표적 세포의 비율은 5:1 이다. 모든 실험은 반복으로 수행되었다. (B) 막대 그래프는 CD45CAR NK45i-92 세포에 의한 세포 용해의 백분율을 CCRF-CEM과의 공동 배양 분석에서의 대조 GFP NK92 세포와 비교하여 보여준다. 데이터는 평균치 +/- S.D. CD45CAR NK45i-92 세포는 대조 GFP NK-92 세포와 비교하여 CCRF-CEM 세포에 대해 약 70 %의 세포 용해를 보여준다. 이들 데이터는 시험관내 공동 배양 분석에서 CD45CAR NK45i-92 세포가 GFP- 대조 NK-92 세포와 비교하여 CD45를 발현하는 CCRF-CEM 세포를 효과적으로 용해시킨다는 것을 시사한다.
도 32 A-C. Jurkat 세포 (표적: T) 와 GFP- 대조 또는 CD45CAR NK45i-92 세포 (효과기: E)의 공동 배양 분석. 5:1 또는 2:1 (E:T) 비율. 6 시간의 인큐베이션. (A) Jurkat 세포가 CMTMR 세포 추적자 염료에 의해 염색된 후에 유동 세포측정법 분석이 수행되었다. 이들 데이터는 Jurkat 세포가 CD45 양성이고 (왼쪽 패널) 대부분은 CD56 음성 세포 (오른쪽 패널)임을 보여준다. (B) Jurkat 세포 (표적: T) 와 대조 또는 CD45CAR NK45i-92 세포 (효과기: E)의 공동 배양 분석의 유동세포측정법 분석. 공동 배양 분석은 5:1 또는 2:1 (E : T) 비율에서 수행되었다. 왼쪽 패널은 5:1 (E:T) 비율에서의 대조 GFP 또는 CD45CAR/CD45KD NK-92 세포와의 공동 배양을 보여주었고 오른쪽 패널은 2:1 (E:T) 비율에서의 대조 GFP 또는 CD45CAR NK45i-92 세포와의 공동배양을 나타내었다. 패널에서의 파란색 점은 남아있는 표적 Jurkat 세포를 나타내고 빨간색 점은 공동 배양 분석에 의한 효과기 세포를 나타낸다. 모든 인큐베이션 시간은 6 시간이었다. 모든 실험은 반복으로 수행되었다. (C) 막대 그래프는 5:1 또는 2:1 (E:T) 비율에서의 CD45CAR NK45i-92 세포에 의한 세포 용해 백분율을 대조 GFP NK92 세포와 비교하여 보여준다. 데이터는 평균치 +/- S.D. 이다. CD45CAR NK45i-92 세포는 두 조건 모두에서 대조 GFP NK-92 세포와 비교하여 Jurkat 세포에 대해 약 60%의 세포 용해를 보여준다. 이 데이터는 CD45CAR NK45i-92 세포가 시험관내 공동 배양 분석에서 GFP- 대조 NK-92 세포와 비교하여 세포 표면 상에서 CD45를 발현하는 Jurkat 세포를 효과적으로 용해시킨다는 것을 시사한다.
도 33A-C. GFP-NK-92 세포 (표적: T) 와 비형질 도입된 NK-92 세포 또는 CD45CAR NK45i-92 세포 (효과기: E)의 공동 배양 분석. 5:1 또는 2:1 (E:T) 비율. 6 시간의 인큐베이션. (A) GFP 대조 NK-92 세포를 사용하여 유동 세포측정법 분석을 수행하였다. 이들 데이터는 GFP 대조 NK-92 세포가 약 99%의 GFP 양성 세포 (녹색 점들)임을 증명한다. (B) GFP 대조 NK-92 세포 (표적: T) 와 비형질 도입된 또는 CD45CAR NK45i-92 세포 (효과기: E)의 공동 배양 측정의 유동 세포측정법 분석. 공동 배양 분석은 5:1 또는 2:1 (E:T) 비율에서 수행되었다. 왼쪽 패널은 비형질 도입된 또는 CD45CAR NK45i-92 세포와의 5:1 (E:T) 비율에서의 공동 배양을 보여주었고 오른쪽 패널은 비형질도입된 또는 CD45CAR NK45i-92 세포와의 2:1 (E:T) 비율에서의 공동배양을 나타냈다. 패널의 녹색 점은 남아있는 표적 GFP NK-92 세포를 나타내고 붉은 점은 공동 배양 분석에 의한 효과기 세포를 나타낸다. 인큐베이션 시간은 6 시간이었다. 모든 실험은 반복으로 수행되었다. (C) 막대 그래프는 5:1 또는 2:1 (E:T) 비율에서의 CD45CAR NK45i-92 세포에 의한 GFP NK-92 세포의 세포 용해 백분율을 비형질 도입된 NK-92 세포와 비교하여 보여준다. 데이터는 평균치 +/- S.D. 이다. CD45CAR NK45i-92 세포는 비형질 도입된 NK-92 세포와 비교하여 GFP NK-92 세포에 대해 2:1 (E:T) 비율에서 약 20 % 세포 용해 및 5:1 (E:T) 비율에서 약 55% 세포 용해를 보여준다. 이 데이터는 CD45CAR NK45i-92 세포가 시험관내 공동 배양 분석에서 비형질 도입된 NK-92 세포에 비교하여 세포 표면 상에서 CD45를 발현하는 GFP NK-92 세포를 효과적으로 용해시킨다는 것을 시사한다. 녹색 점은 각 패널의 오른쪽 상단 4분면에 있다.
도 33D-E. NK45i-92 세포로의 CD45b-BB 또는 CD45b-28의 형질도입 및 CD45b-BB 또는 CD45b-28 형질 도입된 NK45i-92 세포의 세포 분류. (D) CD45b-BB 또는 CD45b-28 렌티바이러스의 NK45i-92 세포로의 형질 도입 후, NK45i-92 상에서의 CD45b-BB CAR 또는 CD45b-28 CAR의 표면 발현 수준은 유동 세포측정법 분석 (중간 패널에서 청색 동그라미로 표시)에 의하여 NK45i-92 세포 (좌측 패널) 와 비교하여 측정되었다. (E) CD45b-BB 또는 CD45b-28 CAR을 발현하는 NK45i-92 세포를 유동 세포측정법 분석으로 분류하였다. 세포 표면 상에서 유동 세포측정법 분석에 의해 약 74%의 CD45b-BB CAR 발현 또는 약 82%의 CD45b-28 CAR 발현이 검출되었다.
도 33 F-G. REH 세포 (표적: T) 와 GFP NK-92 세포 또는 CD45CAR NK45i-92 세포 또는 CD45b-BB NK45i-92 세포 또는 CD45b-28 NK45i-92 세포 (효과기: E)의 공동 배양 분석. 5:1 (E:T) 비율. 20 시간의 인큐베이션. (F) REH 세포 단독 (왼쪽 패널), REH 세포와 대조 GFP 형질 도입된 NK-92 세포의 공동 배양 (2 번째 왼쪽 패널), REH 세포와 CD45CAR NK45i-92 세포의 공동 배양 (중간 패널), REH 세포와 CD45b-BB NK45i-92 세포의 공동 배양 (왼쪽에서 4 번째 패널) 또는 REH 세포와 CD45b-28 NK45i-92 세포의 공동 배양 (오른쪽 패널)의 유동세포측정법 분석. 모든 패널의 파란색 점은 남아있는 표적 REH 세포를 나타내고 빨간색 점은 공동 배양 분석에 의한 효과기 GFP 또는 CARs-NK-92 세포를 나타낸다. REH는 B 급성 림프모구 세포주이다. 모든 인큐베이션 시간은 20 시간이었고 효과 NK 세포: 표적 세포의 비율은 5:1이다. 모든 실험은 반복으로 수행되었다. (G) 막대 그래프는 REH 세포와의 공동 배양 분석에서의 CD45CAR NK45i-92 세포, CD45b-BB NK45i-92 세포 또는 CD45b-28 NK45i-92 세포에 의한 세포 용해의 백분율을 대조 GFP NK92 세포과 비교하여 보여준다. 데이터는 평균치 +/- S.D. 이다. 대조 GFP NK-92 세포와 비교하여, REH 세포에 대해 CD45CAR NK45i-92 세포는 약 76%의 세포 용해를 나타내고 CD45b-BB NK45i-92 세포는 약 79%의 세포 용해를 나타내며 그리고 CD45b-28 NK45i-92는 100 % 세포 용해를 나타낸다. 이들 데이터는 세 종류의 CD45CAR 모두가 REH 세포를 효과적으로 용해시킨다는 것을 시사한다.
도 34 A-B. 구축물 및 T 또는 NK 세포에서의 그의 발현을 설명하기 위한 개략도. (A) CAR (제3 세대), 스시 (sushi)/ IL-15의 조합물은 발현 벡터 상에 조립되고 또 그들의 발현은 SFFV 프로모터에 의해 유도된다. CAR은 스시 (sushi)/IL-15와 함께 P2A 절단 서열과 연결되어 있다. 스시 (sushi)/IL-15 부분은 스시 (sushi) 도메인에 융합된 IL-2 신호 펩타이드로 구성되고 26-아미노산 폴리-프롤린 링커를 통해 IL-5에 연결된다. (B) CAR 및 스시 (sushi)/IL15는 T 또는 NK 세포 상에 존재한다.
도 35 A-B. CD4IL15RA-CAR 발현. (A) HEK-293FT 세포를 GFP (레인 1) 및 CD4IL15RA CAR (레인 2) 및 양성 대조, CD4CAR (레인 3)을 위한 렌티바이러스 플라스미드로 형질감염시켰다. 형질감염 48 시간 후, 상청액을 제거하였고 마우스 항-인간 CD3z 항체와의 웨스턴 블롯을 위해 세포를 또한 분리하였다. (B) HEK-293 세포를 형질 감염된 HEK-293FT 세포 유래의 GFP (왼쪽) 또는 CD4IL15RA-CAR (오른쪽) 바이러스 상청액으로 형질도입시켰다. 3일 동안 인큐베이션한 후, 세포를 수거하고 염소-항-마우스 F(Ab')2로 염색하고 유동 세포측정법으로 분석하였다.
도 36. NK 세포의 CD4IL15RACAR로의 형질도입. NK-92 세포를 형질 감염된 HEK-293FT 세포 유래의 GFP (왼쪽) 또는 CD4IL15RACAR (오른쪽) 바이러스 상청액으로 형질도입시켰다. 두번째 형질도입은 첫번째 형질도입 24시간 후에 수행되었다. 두번째 형질도입 24시간 후에, 세포를 수거하고, 세척하고, 신선한 배지 및 IL-2를 포함하는 조직 배양 플레이트로 옮겼다. 3일 동안 인큐베이션한 후, 세포를 수거하고 염소-항-마우스 F(Ab')2 항체 또는 염소 IgG (대조)로 1:250에서 30 분 동안 염색하였다. 세포를 세척하고 스트렙타비딘(streptavidin)-PE 접합체로 1: 500에서 염색하고, 세척하고, 2% 포르말린에 현탁시키고, 유동 세포측정법으로 분석하였다.
도 37. CD4IL15RACAR을 이용한 T 세포의 형질도입. 왼쪽은 웨스턴 블롯이다. HEK-293FT 세포를 GFP (레인 1) 및 CD4IL15RA-CAR (레인 2)을 위한 렌티바이러스 플라스미드로 형질감염시켰다. 형질감염 48시간 후에, 상청액을 제거하고 그리고 마우스 항-인간 CD3 제타 항체와의 웨스턴 블롯을 위해 세포를 또한 수집하였다. 오른쪽은 CD4IL15RACAR 발현이다. 제대혈 백혈구연층으로부터의 활성화된 T 세포를 형질감염 된 HEK-293FT 세포 유래의 GFP (왼쪽) 또는 CD4IL15RACAR (오른쪽) 바이러스 상청액으로 형질도입하였다. 두번째 형질도입은 첫번째 형질도입 24 시간 후에 수행되었다. 두번째 형질도입 24시간 후에, 세포를 수거하고, 세척하고, 신선한 배지 및 IL-2를 포함하는 조직 배양 플레이트에 옮겼다. 3일 동안 인큐베이션한 후, 세포를 수거하고 염소-항-마우스 F(Ab')2 또는 동형 대조로 30분 동안 염색하였다. GFP (왼쪽) 또는 CD4IL15RA (오른쪽)로 형질 도입되었다. 세포를 세척하고 스트렙타비딘-PE 접합체로 1:250에서 염색하고, 세척하고, 2% 포르말린에 현탁시키고, 유동 세포측정법으로 분석하였다.
도 38 A-B. CD4CAR NK-92 세포와 CD4IL15RA CAR NK-92 세포는 공동 배양에서 KARPAS 299 T 백혈병 세포를 제거한다. (A) GFP 대조 (오른쪽 위), CD4CAR (왼쪽 아래) 또는 CD4IL15RA (오른쪽 아래) 렌티바이러스 상청액으로 형질 도입된 NK-92 세포를 KARPAS 299 세포와 함께 5:1의 비율로 배양하였다. 4시간의 공동 배양 후 세포를 마우스-항-인간 CD4 (APC) 및 CD3 (PerCp) 항체로 염색하고 유동세포측정법 (N = 2)으로 분석하였다. 왼쪽 상단 패널은 표지된 Karpas 299 세포만 보여준다. (B) 용해된 표적 세포의 백분율을 그래프로 나타냈다.
도 39. CD4CAR NK-92 세포 및 CD4IL15RA CAR NK-92 세포는 공동 배양에서 CD4를 발현하는 MOLT4 T 백혈병 세포를 제거한다. GFP 대조 (왼쪽), CD4CAR (가운데), 또는 CD4IL15RA (오른쪽에서 두 번째) 렌티바이러스 상청액으로 형질 도입된 NK-92 세포를 MOLT4 세포와 함께 1:1 또는 2:1의 효과기: 표적 비율로 배양하였다. 하룻밤 동안 공동 배양한 후, 세포를 마우스-항-인간 CD4 (APC) 및 CD56 (PerCp) 항체로 염색하고 유동 세포측정법 (N=2)으로 분석하였다. 오른쪽 상단 패널은 표지된 MOLT4 세포만 보여준다. 용해된 표적 세포의 백분율이 그래프에 표시된다.
도40. CD4IL15RACAR T 세포는 CD4CAR보다 생체 내에서 더 강력한 항-백혈병 효과를 나타낸다. NSG 마우스를 준치사적으로 방사선 조사하고 다음날 루시페레이스-발현하는 MOLM13 세포를 정맥내 (꼬리 정맥) 주사하여 측정 가능한 종양 형성을 유도하였다. 3 일 후, 마우스에게 8 x 106의 CD4CAR, 또는 CD4IL15RACAR T 세포 또는 벡터 대조 T 대조 세포를 정맥내로 한번 주사하였다. 제3, 6, 9 및 11 일째에 마우스에게 RediJect D-Luciferin을 피하 주사하여 IVIS 영상을 시행했다.
도 41. 마우스에서의 종양 감소 백분율을 측정하고 도 40으로부터의 연구에 기초하여 세 그룹간을 비교하였다. CD4CAR 및 CD4IL15RACAR T 주사된 마우스에 대해 측정한 평균 광 강도를 벡터 대조 T 주사 된 마우스의 것과 비교하였고, 잔류 종양 부담과 관련시켰다. 두개의 각 세트에서 CD4CAR T가 왼쪽에 있고 CD4IL15RA CAR T가 오른쪽에 있다.
도 42. HEK 293 세포를 6웰 조직 배양 플레이트에 10% FBS를 함유하는 DMEM 중에서, 표시된 용량을 사용하여 EF1-GFP 또는 SFFV-GFP 바이러스 상청액으로 형질 도입시켰다. 배양 배지는 다음날 아침에 바꿨다. 48시간 후, 형질 도입된 세포를 EVOS 형광 현미경상에서 GFP를 사용하여 10배로 시각화하였다.
도 43. 이전 도면으로부터의 용량을 사용하여 EF1-GFP 또는 SFFV-GFP 바이러스 상청액으로 형질 도입된 HEK 293 세포를 트립신처리하고 포르말린에 현탁시키고, GFP+ 세포의 백분율을 측정하기 위해 FITC 채널을 사용하여 유동 세포측정법 분석을 하였다.
도 44 A-B. EF1-GFP 또는 SFFV-GFP의 바이러스 상청액으로 형질 도입된 활성화된 제대혈 백혈구연층 T 세포를 적은 또는 많은 양의 바이러스 상청액과 함께 트립신처리하고, 포르말린에 현탁시키고, 형질 도입 후의 제 7, 14, 21 및 28 일째의 GFP+ 세포의 비율을 측정하기 위해 FITC 채널을 사용하여 유동 세포측정법 분석을 하였다. (A) 적은 또는 많은 양의 상청액으로 형질 도입된 세포에 대한 GFP+ T 세포 백분율. (B) 보다 적은 양의 SFFV-GFP 상청액으로 형질 도입된 T 세포에서의 GFP+ 세포 백분율과 비교한 높은 양의 EF1-GFP 상청액으로 형질 도입된 GFP+ T 세포의 백분율. (50 μL의 SFFV-GFP 및 1 mL의 EF1-GFP 상청액을 사용하였다). (N=2).
도 45. 악성 형질 세포에서의 리간드 수용체 상호 작용. APRIL 리간드는 TAC1 또는 BCMA와 결합한다. BAFF 리간드는 TAC1, BCMA 또는 BAFF-R와 결합한다.
도 2A. cCAR-T 구축물의 도식적 표현. 상기 구축물은 P2A 펩타이드에 의해 연결된 CAR의 다중 모듈 단위의 발현을 유도하는 SFFV 프로모터를 포함한다. 링커가 절단되면, cCAR들은 분리되고 CD33 및/또는 CD123을 발현하는 표적에 결합한다. 새로운 cCAR 구축물로서, 이 구축물의 활성화 도메인은 CD33 CAR 단편 상의 4-1BB 및 CD123 CAR 상의 CD28 영역을 포함할 수 있으나, 이에 한정되는 것은 아니다.
도 2B. 형질도입된 CD33CD123 cCAR-T 세포의 발현을 도시하는 웨스턴 블롯. 이 도면은 2 개의 상이한 CAR 단백질, 즉, CD33 CAR 및 CD123 CAR의 발현을 도시한다. 링커가 절단되면 CD33 및 CD123 CAR 모두를 발현하는 cCAR-T 세포는 두 개의 명확하고 일관되게 색이 매우 짙은 단백질 밴드를 생성한다. 녹색 형광 단백질 (GFP)은 음성 대조군으로서 포함된다.
도 2C. 형질도입의 효율을 나타내는 유동 세포측정법. 상단 패널은 UCB (제대혈) 및 PB (말초 혈액) T 세포에 사용하기 전 최대 형질 도입 효율을 측정하기 위해 293FT HEK (인간 배아 신장) 세포 상에서 시험된 CD33CD123 cCAR (CD33CD123-2G-CAR라고도 지칭됨)에 대한 렌티바이러스의 역가를 보여준다. 하단 패널은 CD33CD123 cCAR 구축물을 포함하는 렌티바이러스 벡터로 형질 도입된 CD33CD123 cCAR (CD33CD123-2G-CAR 라고도 지칭됨) T- 세포, 및 대조군으로서 GFP- 형질 도입된 세포를 보여준다. 노란색 원에 의해 표시되는 백분율은 형질 도입 효율에 대한 프록시이다.
도 3. 고효율 화합물 CAR (cCAR) 생성 방법을 보여주는 도식.
도 4. CD33CD123-2G CAR-T 세포 (cCAR)의 전골수구성 백혈병 세포주 HL60와의 인큐베이션을 나타내는 공동 배양 분석. cCAR-T 세포 (하부 패널)는 대조 GFP 형질 도입된 T 세포 (상부 패널)와 비교된다. 살상의 효능은 약 24 시간 동안 배양한 후에 남겨진 CD33+ 세포의 집단에 의해 측정된다 (노란색 원으로 둘러싸임).
도 5. cCAR-T 세포의 약 100% CD33과 약 50-80% CD123을 발현하는 골수성 백혈병 세포주 KG-1a와의 인큐베이션을 나타내는 공동 배양 분석. cCAR-T 세포 (하부 패널)는 대조 GFP 형질 도입된 T 세포 (상부 패널)와 비교된다. 살상의 효능은 약 24시간 동안 배양한 후에 남겨진 CD33+ 세포의 집단에 의해 측정된다.
도 6. cCAR-T 세포의 AML 환자 샘플 (여기서는 AML-9라고 지칭됨)과의 인큐베이션을 나타내는 공동 배양 분석. 환자 세포는 예를 들어 백혈병 세포, 단핵구 및 다른 유형의 모세포와 같은 혼합된 세포 집단을 포함한다. CD33은 백혈병 세포에 대한 특이적 마커인 CD34와 마찬가지로 CAR-T 작용에 대한 마커로서 또한 작용한다. CAR-T 패널 (우측)은 대조 GFP 형질 도입된 T 세포 (중간)와 비교된다. 살상의 효능은 적어도 24시간 동안 인큐베이션한 후에 남겨진 CD33+/CD34+ 세포의 집단에 의해 측정된다.
도 7. cCAR-T 세포의 B-ALL 환자 샘플 (여기서는 Sp-BM-B6이라고 지칭됨)과의 인큐베이션을 나타내는 공동 배양 분석. 환자 세포는 예를 들어, 백혈병 세포, 단핵구 및 다른 유형의 모세포와 같은 혼합된 세포 집단을 포함한다. CD34는 백혈병 세포에 대한 특이적 마커로서 작용한다. CAR-T 패널 (우측)은 대조 GFP 형질 도입된 T 세포 (중간)와 비교된다. 살상의 효능은 적어도 24시간 동안 인큐베이션한 후에 남겨진 CD34+ 세포 집단에 의해 측정된다.
도 8. NK-92 세포에서의 CD33CD123 cCAR 발현. CD33CD123 cCAR 발현은 염소-항-마우스 항체, F(ab)2를 사용하여 검출된다.
도 9. cCAR NK-92 세포의 HL-60과의 인큐베이션을 나타내는 공동 배양 분석. cCAR NK-92 세포는 GFP 형질 도입된 NK-92 세포와 비교된다. 살상의 효능은 약 24시간 동안 인큐베이션한 후 남은 CD33+ 세포의 집단에 의해 측정된다.
도 10. cCAR NK-92 세포의 KG1a와의 인큐베이션을 나타내는 공동 배양 분석. cCAR NK 세포 패널은 GFP 형질 도입된 NK-92 세포와 비교된다. 살상의 효능은 약 24시간 동안 인큐베이션한 후 남은 CD33+ 세포의 집단에 의해 측정된다.
도 11. HL-60 또는 KG1a을 사용한 CD33CD123 cCAR (CAR-CD33/123) NK-92 세포의 용량 반응.
살상의 효능은 약 24시간 동안 인큐베이션한 후 남은 CD33+ 세포의 집단에 의해 측정된다.
도 12. KG11 세포의 두 집단에서의 CD33CD123 cCAR NK-92 세포의 살상 능력과 대조의 비교. CAR-CD33/123 (CD33CD123 cCAR NK-92 세포) 와 표적 세포 kG1a의 상이한 비율로 분석을 수행하였다. 살상의 효능은 약 24시간 동안 인큐베이션한 후 남은 CD33+CD123+ 또는 CD33+CD123- 세포의 집단에 의해 측정된다.
도 13. cCAR의 도식적 표현. 구축물은 링커에 의해 연결된 CAR의 다중 모듈 단위의 발현을 유도하는 SFFV 프로모터를 포함한다. 링커가 절단되면, cCAR들은 분리되고 CD19 및/또는 CD20, 및/또는 CD22 및/또는 138의 다양한 표적 항원의 조합을 발현하는 표적에 결합한다. 다중 cCAR은, 예를 들어 4-1BB (4-BB로도 표기함) 및/또는 CD28로 제한하는 것 없이, 동일하거나 또는 상이한 보조 자극 도메인을 이용한다.
도 14 A-C. BCMA-CS1 cCAR 구축물 도식 (BC1cCAR). (A) 구축물은 P2A 펩타이드에 의해 연결된 CAR의 2 개의 모듈 단위의 발현을 유도하는 SFFV 프로모터로 구성된다. 이 P2A 펩티드가 절단되면, cCAR들은 분리되고 BCMA 및/또는 CS1을 발현하는 표적에 결합한다. 2개의 CAR 단위는 동일한 보조 자극 도메인 4-1BB를 사용한다. (B) 벡터 (왼쪽)와 BC1cCAR (오른쪽, 사각형으로 강조됨)에 대한 T 세포 표면에서의 BC1cCAR 발현의 유동 세포측정법 분석이며, BC1cCAR은 F(Ab)2에 대해 15.3 %의 양성을 보임. 동형 대조에 대해 게이팅함. (C) BCMA cDNA (BCMA-K562) (NIH, Kochenderfer로부터 얻음)로 형질 도입한 K562 세포와의 공동 배양으로 BC1cCAR-T 세포의 예비 기능 검증. 막대 그래프는 대조 T 세포에 대한 BCMA-K562 세포주의 용해 그리고 대조에 대한 야생형 K562 (wt-K562)의 용해를 보여준다.
도 14D. 각 단위에 대해 4-1BB 또는 CD28의 2개의 상이한 보조 자극 도메인을 사용하는 BCMA-CS1-2G 구축물. 이 구축물은 P2A 펩타이드에 의해 연결된 CAR의 2개의 모듈 단위의 발현을 유도하는 SFFV 프로모터를 포함한다. 이 P2A 펩타이드가 절단되면, cCAR들은 분리되고 BCMA 및/또는 CS1을 발현하는 표적에 결합한다. 2개의 CAR 단위는 4-1BB 또는 CD28의 서로 다른 보조 자극 도메인을 사용한다. 벡터 (왼쪽)와 BC1cCAR (오른쪽, 사각형으로 강조됨)에 대한 T 세포 표면에서의 BC1cCAR 발현의 유동 세포측정법 분석이며, BC1cCAR은 F(Ab)2에 대해 희귀한 양성 세포를 보임. 동형 대조에 대해 게이팅함.
도 14E. HEK-293FT 세포에서의 BC1cCAR 및 BCMA-CS1-2G의 단백질 발현. HEK-293FT 세포를 GFP (레인 1), BC1cCAR (레인 2), CD269-CS1-2G (레인 3)를 위한 렌티바이러스 플라스미드로 형질 감염시키고, 형질 감염 48시간 후에 상청액을 제거하고 세포를 또한 분리하였다. 웨스턴 블롯을 위하여 세포를 용해하고 마우스 항-인간 CD3z 항체로 프로브하였다.
도 15A-B. MM1S 세포주 공동배양. 공동배양을 24시간 이내에 수행하고 수집하고 유동 세포측정법을 통해 분석하였다. 표적 MM1S 세포 (골수종 세포)를 Cytotracker (CMTMR) 염료로 표지하여 효과 T 세포로부터 구별하였다. 항 BCMA (CD269) 및 항-CS1 (CD319) 항체로 집단을 게이팅하였다. 도 15A: 공동 배양의 유동 세포측정법 도시. 도 15B: 오른쪽: 용해 대 E:T 비율의 그래픽 요약.
도 16A-B. RPMI-8226 세포주 공동 배양. 공동 배양을 24시간 이내에 수행하고 수집하고 유동 세포측정법을 통해 분석하였다. 표적 RPMI-8226 세포를 Cytotracker (CMTMR) 염료로 표지하여 효과 T 세포로부터 구별하였다. 항-BCMA (CD269) 및 항-CS1 (CD319) 항체로 집단을 게이팅하였다. 도 16A: 공동 배양의 유동 세포측정법 묘사. 도 16B: 용해 대 E:T 비율의 그래픽 요약.
도17 A-B. U266 세포주 공동 배양. 공동 배양을 24 시간 이내에 수행하고 수집하고 유동 세포측정법을 통해 분석하였다. 표적 U266 세포를 Cytotracker (CMTMR) 염료로 표지하여 효과 T 세포로부터 구별하였다. 항 BCMA (CD269) 및 항-CS1 (CD319) 항체로 집단을 게이팅하였다. (A) 공동 배양의 유동 세포측정법 도시. (B) 용해 대 E:T 비율의 그래픽 요약.
도 18A-B. MM10-G 원발성 환자 샘플 공동 배양 및 특이적 용해. 공동 배양을 24시간 이내에 수행하고 수집하고 유동세포측정법을 통해 분석하였다. 표적 MM10-G 세포를 Cytotracker (CMTMR) 염료로 표지하여 효과 T 세포로부터 구별하였다. 항 BCMA (CD269) 및 항-CS1 (CD319) 항체로 집단을 게이팅하였다. 특히, 게이팅은 MM10-G가 뚜렷한 BCMA+ 및 CS1+ 집단을 나타냄을 보여준다. 도 18A: 공동 배양의 유동 세포측정법 도시. 도 18B: 용해 대 E:T 비율의 그래픽 요약.
도 19A-B. MM7-G 원발성 환자 샘플 공동 배양 및 특이적 용해. 공동 배양을 24시간 이내에 수행하고 수집하고 유동 세포측정법을 통해 분석하였다. 표적 MM7-G 세포를 Cytotracker (CMTMR) 염료로 표지하여 효과 T 세포로부터 구별하였다. 항-BCMA (CD269) 및 항-CS1 (CD319) 항체로 집단을 게이팅하였다. 도 19A: 공동 배양의 유동 세포측정법 도시. 도 19B: 용해 대 E:T 비율의 그래픽 요약.
도 20A-B. MM11-G 원발성 환자 샘플 공동 배양 및 특이적 용해. 공동 배양을 24 시간 이내에 수행하고 수집하고 유동 세포측정법을 통해 분석하였다. 표적 MM11-G 세포를 Cytotracker (CMTMR) 염료로 표지하여 효과 T 세포로부터 구별하였다. 항-BCMA (CD269) 및 항-CS1 (CD319) 항체로 집단을 게이팅하였다. 도 20A: 공동 배양의 유동세포측정법 도시. 도 20B: 용해 대 E:T 비율의 그래픽 요약.
도 21. CD269-CS1-BBCAR NK 세포는 생체내에서 항-백혈병 효과를 나타낸다. NSG 마우스에게 준치사적 방사선을 조사하고 다음 날 루시페레이스를 발현하는 MM.1S 다발성 골수종 세포를 정맥내 주사하여 측정 가능한 종양 형성을 유도하였다. 3 일 후, 마우스에게 8 x 106 CD269-CS1-BBCAR NK 세포 또는 벡터 대조 NK 대조 세포를 정맥내 주사하였다. 제 3, 6, 및 8 일에 마우스에게 RediJect D-Luciferin을 피하 주사하여 IVIS 영상을 시행하였다. CD269-CS1-BBCAR NK 주사된 마우스에 대해 측정된 평균 광 강도를 벡터 대조 NK 주사된 마우스의 것과 비교하였다.
도 22. 도 21의 연구에 근거하여 마우스의 생존율을 측정하고 두 그룹 사이에서의 마우스의 생존율을 비교하였다.
도 23. CRISPR / Cas9 간섭 시스템. sgRNA 및 Cas9 퓨로마이신의 발현은 U6 및 SFFV 프로모터에 의해 각각 유도된다. Cas9는 E2A 자가절단 서열에 의해 퓨로마이신 내성유전자와 연결되어 있다.
도 24. 혈액 악성 종양을 표적으로하는 CAR T 또는 NK 세포의 생성을 위한 단계의 예를 제공하는 도식.
도 25. CRISPR/Cas9 렌티바이러스 시스템을 사용한 안정된 CD45 녹다운 NK-92 세포의 생성 및 세포 분류. 유동 세포측정법 분석은 NK-92 세포 표면 상에서의 CD45 발현 수준을 나타내었다 (왼쪽 패널). sgCD45B CRISPR을 NK-92 세포에 형질도입한 후, 형질도입된 세포를 퓨로마이신을 함유하는 배지 중에서 수주 동안 배양하였다. CD45 음성 NK-92 세포를 CD45 항체를 사용하여 결정하고 분류하였다. 안정된 NK45i-92 (CD45 녹다운) NK-92 세포의 순도는 유동 세포측정법 분석에 의해 측정되었다 (오른쪽 패널). 이 데이터는 NK45i-92 세포가 성공적으로 생성되고 얻어졌다는 것을 보여 주었다.
도 26. 야생형, GFP 형질도입된 NK-92 또는 NK45i-92NK 세포의 세포 성장 곡선. NK-92 세포에서의 CD45- 녹다운 (KD)에 의해 야기된 세포 증식에 대한 효과를 평가하기 위해, 24 웰 플레이트 (well plates)에 시딩한 후 48 시간 및 96 시간에 NK-92 (●), GFP-형질도입된 NK-92 (■) 및 NK45i-92 (▲)의 세포 수를 계수하였다. 48시간 시점에 IL-2를 첨가하였다 (n=3회의 독립적인 실험을 반복으로 수행하였다). 데이터는 평균치+/- S.D. 이다. 이들 데이터는 NK-92상의 CD45 수용체의 녹다운이 비형질도입된 NK-92 또는 GFP-형질도입된 NK-92 세포와 비교하여 유사한 세포 성장 곡선을 보인다는 것을 나타냈다.
도 27 A-B. CCRF-CEM (표적: T) 및 GFP NK-92 또는 GFP NK45i-92 세포 (효과기: E), 5:1 (E:T) 비율의 공동 배양 분석. 16시간 인큐베이션. (A) CCRF-CEM 단독 (좌측 패널의 파란색 점), CCRF-CEM과 대조 GFP 형질 도입된 NK-92 세포의 공동 배양 (중간 패널) 또는 CCRF-CEM과 GFP NK45i-92 세포의 공동 배양 (우측 패널)의 유동 세포측정법 분석. 모든 패널에서의 파란색 점은 남아있는 표적 CCRF-CEM 세포를 나타내고 빨간색 점은 공동 배양 분석에 의한 효과 세포를 나타낸다. 인큐베이션 시간은 전부 16 시간이었고 효과 T 세포: 표적 세포의 비율은 5:1이었다. 모든 실험은 반복으로 수행되었다. (B) 막대 그래프는 CCRF-CEM과의 공동 배양 분석에서 GFP 형질도입된 NK45i-92 세포에 의한 세포 용해의 백분율을 대조 GFP 형질도입된 NK92 세포와 비교하여 보여준다. 이들 데이터는 시험관내 공동 배양 분석에서 NK-92 세포에서의 CD45의 녹다운이 GFP-대조 NK-92 세포에 비교하여 CCRF-CEM 세포에 대한 살상 활성의 유의한 차이를 나타내지 않는다는 것을 시사한다. 파란색 점은 왼쪽 상단 4분면에 있다.
도 28 A-B. CCRF-CEM (표적: T) 그리고 GFP NK-92, CD5CAR NK-92 또는 CD5CAR NK45i-92 세포 (효과기: E)와의 공동 배양 분석. 5: 1 (E: T) 비율. 16 시간 인큐베이션. (A) 오른쪽에서 왼쪽으로, CCRF-CEM 단독 (왼쪽 패널), CCRF-CEM과 대조 GFP NK-92 세포의 공동 배양 (중간 왼쪽 패널), CCRF-CEM과 CD5CAR NK-92 세포의 공동 배양 (중간 오른쪽 패널), CCRF-CEM과 CD5CAR NK45i-92 세포의 공동 배양 (오른쪽 패널)의 유동 세포측정법 분석. 모든 패널의 파란색 점은 남아있는 표적 CCRF-CEM 세포를 나타내고 빨간색 점은 공동 배양 분석에 의한 효과 세포를 나타낸다. 인큐베이션 시간은 전부 16시간이었고 효과 T 세포: 표적 세포의 비율은 5:1이었다. 모든 실험은 반복으로 수행되었다. (B) 막대 그래프는 CCRF-CEM과의 공동 배양 분석에서 CD5CAR NK-92 세포 또는 CD5CAR NK45i-92 세포에 의한 세포 용해의 백분율을 대조 GFP NK92 세포와 비교하여 보여준다. 데이터는 평균치 +/- S.D. 이다. CD5CAR NK 세포 및 CD5CAR NK45i-92 세포 모두 대조 GFP NK-92 세포와 비교하여 CD5-양성 CCRF-CEM에 대해 100%에 가까운 세포 살상 활성을 나타낸다. 이들 데이터는 CD5CAR NK 세포 및 CD5CAR NK45i-92 세포가 GFP-대조 NK-92 세포와 비교하여 시험관내 공동 배양 분석에서 CD5를 발현하는 CCRF-CEM 세포를 효과적으로 용해시킬 수 있고, CD45의 녹다운이 NK-92 세포에서의 살상 활성을 위한 세포 기능에 영향을 미치지 않는다는 증거를 제공한다. 파란색 점은 왼쪽으로부터 시작하여 처음 두 패널의 왼쪽 상단 4분면에 있다.
도 29A-B. CD45CAR 구축물의 구성과 그 발현. (A) CD45CAR 렌티바이러스 벡터의 도식적 표시. CD45CAR 구축물은 모듈화된 신호전달 도메인이고 리더 서열, 항-CD45scFv, 힌지 도메인 (H), 막통과 도메인 (TM), 구축물의 제3세대 CAR로서의 특징인 두 개의 보조 자극 도메인 (CD28 및 4-1BB), 및 세포내 신호전달 도메인 CD3 제타를 포함한다. (B), HEK-293FT 세포를 GFP (레인 1) 및 CD45CAR (레인 2)를 위한 렌티바이러스 플라스미드로 형질 감염시켰다. 형질 감염 48시간 후에 상청액을 제거하고 세포도 분리하였다. 웨스턴 블롯을 위해 세포를 용해시키고 마우스 항-인간 CD3z 항체로 프로브하였다.
도 30 A-B. NK45i-92 세포로의 CD45CAR의 형질도입 및 CD45CAR 형질도입된 세포의 세포분류. (A) CD45CAR 렌티바이러스가 NK45i-92 세포로 형질도입된 후에 NK45i-92상의 CD45CAR의 발현 수준을 유동 세포측정법 분석 (중간 패널에서 파란 동그라미로 표시)으로 NK45i-92 세포 (왼쪽 패널)와 비교하여 측정하였다. CD45CAR 발현된 NK45i-92 세포를 분류하고 세포 표면 상의 CD45 발현 수준을 유동 세포측정법 분석 (오른쪽 패널)에 의해 측정하였다. (B) 유동 세포측정법 분석에 의해 검출된 세포 표면 상 CD45CAR 발현은 약 87%였다.
도 31A-B. CCRF-CEM (표적: T) 와 GFP NK-92 또는 CD45CAR NK45i-92 세포 (효과기: E)와의 공동 배양 분석. 5:1 (E:T) 비율. 16 시간 인큐베이션. (A) CCRF-CEM과 대조 GFP 형질도입된 NK-92 세포의 공동 배양 (왼쪽 패널) 또는 CCRF-CEM과 CD45CAR NK45i-92 세포의 공동 배양 (오른쪽 패널)의 유동 세포측정법 분석. 모든 패널의 파란색 점은 남아있는 표적 CCRF-CEM 세포를 나타내고 빨간색 점은 공동 배양 분석에 의한 효과기 NK-92 세포를 보여준다. 모든 인큐베이션 시간은 16 시간이었고 효과 T세포: 표적 세포의 비율은 5:1 이다. 모든 실험은 반복으로 수행되었다. (B) 막대 그래프는 CD45CAR NK45i-92 세포에 의한 세포 용해의 백분율을 CCRF-CEM과의 공동 배양 분석에서의 대조 GFP NK92 세포와 비교하여 보여준다. 데이터는 평균치 +/- S.D. CD45CAR NK45i-92 세포는 대조 GFP NK-92 세포와 비교하여 CCRF-CEM 세포에 대해 약 70 %의 세포 용해를 보여준다. 이들 데이터는 시험관내 공동 배양 분석에서 CD45CAR NK45i-92 세포가 GFP- 대조 NK-92 세포와 비교하여 CD45를 발현하는 CCRF-CEM 세포를 효과적으로 용해시킨다는 것을 시사한다.
도 32 A-C. Jurkat 세포 (표적: T) 와 GFP- 대조 또는 CD45CAR NK45i-92 세포 (효과기: E)의 공동 배양 분석. 5:1 또는 2:1 (E:T) 비율. 6 시간의 인큐베이션. (A) Jurkat 세포가 CMTMR 세포 추적자 염료에 의해 염색된 후에 유동 세포측정법 분석이 수행되었다. 이들 데이터는 Jurkat 세포가 CD45 양성이고 (왼쪽 패널) 대부분은 CD56 음성 세포 (오른쪽 패널)임을 보여준다. (B) Jurkat 세포 (표적: T) 와 대조 또는 CD45CAR NK45i-92 세포 (효과기: E)의 공동 배양 분석의 유동세포측정법 분석. 공동 배양 분석은 5:1 또는 2:1 (E : T) 비율에서 수행되었다. 왼쪽 패널은 5:1 (E:T) 비율에서의 대조 GFP 또는 CD45CAR/CD45KD NK-92 세포와의 공동 배양을 보여주었고 오른쪽 패널은 2:1 (E:T) 비율에서의 대조 GFP 또는 CD45CAR NK45i-92 세포와의 공동배양을 나타내었다. 패널에서의 파란색 점은 남아있는 표적 Jurkat 세포를 나타내고 빨간색 점은 공동 배양 분석에 의한 효과기 세포를 나타낸다. 모든 인큐베이션 시간은 6 시간이었다. 모든 실험은 반복으로 수행되었다. (C) 막대 그래프는 5:1 또는 2:1 (E:T) 비율에서의 CD45CAR NK45i-92 세포에 의한 세포 용해 백분율을 대조 GFP NK92 세포와 비교하여 보여준다. 데이터는 평균치 +/- S.D. 이다. CD45CAR NK45i-92 세포는 두 조건 모두에서 대조 GFP NK-92 세포와 비교하여 Jurkat 세포에 대해 약 60%의 세포 용해를 보여준다. 이 데이터는 CD45CAR NK45i-92 세포가 시험관내 공동 배양 분석에서 GFP- 대조 NK-92 세포와 비교하여 세포 표면 상에서 CD45를 발현하는 Jurkat 세포를 효과적으로 용해시킨다는 것을 시사한다.
도 33A-C. GFP-NK-92 세포 (표적: T) 와 비형질 도입된 NK-92 세포 또는 CD45CAR NK45i-92 세포 (효과기: E)의 공동 배양 분석. 5:1 또는 2:1 (E:T) 비율. 6 시간의 인큐베이션. (A) GFP 대조 NK-92 세포를 사용하여 유동 세포측정법 분석을 수행하였다. 이들 데이터는 GFP 대조 NK-92 세포가 약 99%의 GFP 양성 세포 (녹색 점들)임을 증명한다. (B) GFP 대조 NK-92 세포 (표적: T) 와 비형질 도입된 또는 CD45CAR NK45i-92 세포 (효과기: E)의 공동 배양 측정의 유동 세포측정법 분석. 공동 배양 분석은 5:1 또는 2:1 (E:T) 비율에서 수행되었다. 왼쪽 패널은 비형질 도입된 또는 CD45CAR NK45i-92 세포와의 5:1 (E:T) 비율에서의 공동 배양을 보여주었고 오른쪽 패널은 비형질도입된 또는 CD45CAR NK45i-92 세포와의 2:1 (E:T) 비율에서의 공동배양을 나타냈다. 패널의 녹색 점은 남아있는 표적 GFP NK-92 세포를 나타내고 붉은 점은 공동 배양 분석에 의한 효과기 세포를 나타낸다. 인큐베이션 시간은 6 시간이었다. 모든 실험은 반복으로 수행되었다. (C) 막대 그래프는 5:1 또는 2:1 (E:T) 비율에서의 CD45CAR NK45i-92 세포에 의한 GFP NK-92 세포의 세포 용해 백분율을 비형질 도입된 NK-92 세포와 비교하여 보여준다. 데이터는 평균치 +/- S.D. 이다. CD45CAR NK45i-92 세포는 비형질 도입된 NK-92 세포와 비교하여 GFP NK-92 세포에 대해 2:1 (E:T) 비율에서 약 20 % 세포 용해 및 5:1 (E:T) 비율에서 약 55% 세포 용해를 보여준다. 이 데이터는 CD45CAR NK45i-92 세포가 시험관내 공동 배양 분석에서 비형질 도입된 NK-92 세포에 비교하여 세포 표면 상에서 CD45를 발현하는 GFP NK-92 세포를 효과적으로 용해시킨다는 것을 시사한다. 녹색 점은 각 패널의 오른쪽 상단 4분면에 있다.
도 33D-E. NK45i-92 세포로의 CD45b-BB 또는 CD45b-28의 형질도입 및 CD45b-BB 또는 CD45b-28 형질 도입된 NK45i-92 세포의 세포 분류. (D) CD45b-BB 또는 CD45b-28 렌티바이러스의 NK45i-92 세포로의 형질 도입 후, NK45i-92 상에서의 CD45b-BB CAR 또는 CD45b-28 CAR의 표면 발현 수준은 유동 세포측정법 분석 (중간 패널에서 청색 동그라미로 표시)에 의하여 NK45i-92 세포 (좌측 패널) 와 비교하여 측정되었다. (E) CD45b-BB 또는 CD45b-28 CAR을 발현하는 NK45i-92 세포를 유동 세포측정법 분석으로 분류하였다. 세포 표면 상에서 유동 세포측정법 분석에 의해 약 74%의 CD45b-BB CAR 발현 또는 약 82%의 CD45b-28 CAR 발현이 검출되었다.
도 33 F-G. REH 세포 (표적: T) 와 GFP NK-92 세포 또는 CD45CAR NK45i-92 세포 또는 CD45b-BB NK45i-92 세포 또는 CD45b-28 NK45i-92 세포 (효과기: E)의 공동 배양 분석. 5:1 (E:T) 비율. 20 시간의 인큐베이션. (F) REH 세포 단독 (왼쪽 패널), REH 세포와 대조 GFP 형질 도입된 NK-92 세포의 공동 배양 (2 번째 왼쪽 패널), REH 세포와 CD45CAR NK45i-92 세포의 공동 배양 (중간 패널), REH 세포와 CD45b-BB NK45i-92 세포의 공동 배양 (왼쪽에서 4 번째 패널) 또는 REH 세포와 CD45b-28 NK45i-92 세포의 공동 배양 (오른쪽 패널)의 유동세포측정법 분석. 모든 패널의 파란색 점은 남아있는 표적 REH 세포를 나타내고 빨간색 점은 공동 배양 분석에 의한 효과기 GFP 또는 CARs-NK-92 세포를 나타낸다. REH는 B 급성 림프모구 세포주이다. 모든 인큐베이션 시간은 20 시간이었고 효과 NK 세포: 표적 세포의 비율은 5:1이다. 모든 실험은 반복으로 수행되었다. (G) 막대 그래프는 REH 세포와의 공동 배양 분석에서의 CD45CAR NK45i-92 세포, CD45b-BB NK45i-92 세포 또는 CD45b-28 NK45i-92 세포에 의한 세포 용해의 백분율을 대조 GFP NK92 세포과 비교하여 보여준다. 데이터는 평균치 +/- S.D. 이다. 대조 GFP NK-92 세포와 비교하여, REH 세포에 대해 CD45CAR NK45i-92 세포는 약 76%의 세포 용해를 나타내고 CD45b-BB NK45i-92 세포는 약 79%의 세포 용해를 나타내며 그리고 CD45b-28 NK45i-92는 100 % 세포 용해를 나타낸다. 이들 데이터는 세 종류의 CD45CAR 모두가 REH 세포를 효과적으로 용해시킨다는 것을 시사한다.
도 34 A-B. 구축물 및 T 또는 NK 세포에서의 그의 발현을 설명하기 위한 개략도. (A) CAR (제3 세대), 스시 (sushi)/ IL-15의 조합물은 발현 벡터 상에 조립되고 또 그들의 발현은 SFFV 프로모터에 의해 유도된다. CAR은 스시 (sushi)/IL-15와 함께 P2A 절단 서열과 연결되어 있다. 스시 (sushi)/IL-15 부분은 스시 (sushi) 도메인에 융합된 IL-2 신호 펩타이드로 구성되고 26-아미노산 폴리-프롤린 링커를 통해 IL-5에 연결된다. (B) CAR 및 스시 (sushi)/IL15는 T 또는 NK 세포 상에 존재한다.
도 35 A-B. CD4IL15RA-CAR 발현. (A) HEK-293FT 세포를 GFP (레인 1) 및 CD4IL15RA CAR (레인 2) 및 양성 대조, CD4CAR (레인 3)을 위한 렌티바이러스 플라스미드로 형질감염시켰다. 형질감염 48 시간 후, 상청액을 제거하였고 마우스 항-인간 CD3z 항체와의 웨스턴 블롯을 위해 세포를 또한 분리하였다. (B) HEK-293 세포를 형질 감염된 HEK-293FT 세포 유래의 GFP (왼쪽) 또는 CD4IL15RA-CAR (오른쪽) 바이러스 상청액으로 형질도입시켰다. 3일 동안 인큐베이션한 후, 세포를 수거하고 염소-항-마우스 F(Ab')2로 염색하고 유동 세포측정법으로 분석하였다.
도 36. NK 세포의 CD4IL15RACAR로의 형질도입. NK-92 세포를 형질 감염된 HEK-293FT 세포 유래의 GFP (왼쪽) 또는 CD4IL15RACAR (오른쪽) 바이러스 상청액으로 형질도입시켰다. 두번째 형질도입은 첫번째 형질도입 24시간 후에 수행되었다. 두번째 형질도입 24시간 후에, 세포를 수거하고, 세척하고, 신선한 배지 및 IL-2를 포함하는 조직 배양 플레이트로 옮겼다. 3일 동안 인큐베이션한 후, 세포를 수거하고 염소-항-마우스 F(Ab')2 항체 또는 염소 IgG (대조)로 1:250에서 30 분 동안 염색하였다. 세포를 세척하고 스트렙타비딘(streptavidin)-PE 접합체로 1: 500에서 염색하고, 세척하고, 2% 포르말린에 현탁시키고, 유동 세포측정법으로 분석하였다.
도 37. CD4IL15RACAR을 이용한 T 세포의 형질도입. 왼쪽은 웨스턴 블롯이다. HEK-293FT 세포를 GFP (레인 1) 및 CD4IL15RA-CAR (레인 2)을 위한 렌티바이러스 플라스미드로 형질감염시켰다. 형질감염 48시간 후에, 상청액을 제거하고 그리고 마우스 항-인간 CD3 제타 항체와의 웨스턴 블롯을 위해 세포를 또한 수집하였다. 오른쪽은 CD4IL15RACAR 발현이다. 제대혈 백혈구연층으로부터의 활성화된 T 세포를 형질감염 된 HEK-293FT 세포 유래의 GFP (왼쪽) 또는 CD4IL15RACAR (오른쪽) 바이러스 상청액으로 형질도입하였다. 두번째 형질도입은 첫번째 형질도입 24 시간 후에 수행되었다. 두번째 형질도입 24시간 후에, 세포를 수거하고, 세척하고, 신선한 배지 및 IL-2를 포함하는 조직 배양 플레이트에 옮겼다. 3일 동안 인큐베이션한 후, 세포를 수거하고 염소-항-마우스 F(Ab')2 또는 동형 대조로 30분 동안 염색하였다. GFP (왼쪽) 또는 CD4IL15RA (오른쪽)로 형질 도입되었다. 세포를 세척하고 스트렙타비딘-PE 접합체로 1:250에서 염색하고, 세척하고, 2% 포르말린에 현탁시키고, 유동 세포측정법으로 분석하였다.
도 38 A-B. CD4CAR NK-92 세포와 CD4IL15RA CAR NK-92 세포는 공동 배양에서 KARPAS 299 T 백혈병 세포를 제거한다. (A) GFP 대조 (오른쪽 위), CD4CAR (왼쪽 아래) 또는 CD4IL15RA (오른쪽 아래) 렌티바이러스 상청액으로 형질 도입된 NK-92 세포를 KARPAS 299 세포와 함께 5:1의 비율로 배양하였다. 4시간의 공동 배양 후 세포를 마우스-항-인간 CD4 (APC) 및 CD3 (PerCp) 항체로 염색하고 유동세포측정법 (N = 2)으로 분석하였다. 왼쪽 상단 패널은 표지된 Karpas 299 세포만 보여준다. (B) 용해된 표적 세포의 백분율을 그래프로 나타냈다.
도 39. CD4CAR NK-92 세포 및 CD4IL15RA CAR NK-92 세포는 공동 배양에서 CD4를 발현하는 MOLT4 T 백혈병 세포를 제거한다. GFP 대조 (왼쪽), CD4CAR (가운데), 또는 CD4IL15RA (오른쪽에서 두 번째) 렌티바이러스 상청액으로 형질 도입된 NK-92 세포를 MOLT4 세포와 함께 1:1 또는 2:1의 효과기: 표적 비율로 배양하였다. 하룻밤 동안 공동 배양한 후, 세포를 마우스-항-인간 CD4 (APC) 및 CD56 (PerCp) 항체로 염색하고 유동 세포측정법 (N=2)으로 분석하였다. 오른쪽 상단 패널은 표지된 MOLT4 세포만 보여준다. 용해된 표적 세포의 백분율이 그래프에 표시된다.
도40. CD4IL15RACAR T 세포는 CD4CAR보다 생체 내에서 더 강력한 항-백혈병 효과를 나타낸다. NSG 마우스를 준치사적으로 방사선 조사하고 다음날 루시페레이스-발현하는 MOLM13 세포를 정맥내 (꼬리 정맥) 주사하여 측정 가능한 종양 형성을 유도하였다. 3 일 후, 마우스에게 8 x 106의 CD4CAR, 또는 CD4IL15RACAR T 세포 또는 벡터 대조 T 대조 세포를 정맥내로 한번 주사하였다. 제3, 6, 9 및 11 일째에 마우스에게 RediJect D-Luciferin을 피하 주사하여 IVIS 영상을 시행했다.
도 41. 마우스에서의 종양 감소 백분율을 측정하고 도 40으로부터의 연구에 기초하여 세 그룹간을 비교하였다. CD4CAR 및 CD4IL15RACAR T 주사된 마우스에 대해 측정한 평균 광 강도를 벡터 대조 T 주사 된 마우스의 것과 비교하였고, 잔류 종양 부담과 관련시켰다. 두개의 각 세트에서 CD4CAR T가 왼쪽에 있고 CD4IL15RA CAR T가 오른쪽에 있다.
도 42. HEK 293 세포를 6웰 조직 배양 플레이트에 10% FBS를 함유하는 DMEM 중에서, 표시된 용량을 사용하여 EF1-GFP 또는 SFFV-GFP 바이러스 상청액으로 형질 도입시켰다. 배양 배지는 다음날 아침에 바꿨다. 48시간 후, 형질 도입된 세포를 EVOS 형광 현미경상에서 GFP를 사용하여 10배로 시각화하였다.
도 43. 이전 도면으로부터의 용량을 사용하여 EF1-GFP 또는 SFFV-GFP 바이러스 상청액으로 형질 도입된 HEK 293 세포를 트립신처리하고 포르말린에 현탁시키고, GFP+ 세포의 백분율을 측정하기 위해 FITC 채널을 사용하여 유동 세포측정법 분석을 하였다.
도 44 A-B. EF1-GFP 또는 SFFV-GFP의 바이러스 상청액으로 형질 도입된 활성화된 제대혈 백혈구연층 T 세포를 적은 또는 많은 양의 바이러스 상청액과 함께 트립신처리하고, 포르말린에 현탁시키고, 형질 도입 후의 제 7, 14, 21 및 28 일째의 GFP+ 세포의 비율을 측정하기 위해 FITC 채널을 사용하여 유동 세포측정법 분석을 하였다. (A) 적은 또는 많은 양의 상청액으로 형질 도입된 세포에 대한 GFP+ T 세포 백분율. (B) 보다 적은 양의 SFFV-GFP 상청액으로 형질 도입된 T 세포에서의 GFP+ 세포 백분율과 비교한 높은 양의 EF1-GFP 상청액으로 형질 도입된 GFP+ T 세포의 백분율. (50 μL의 SFFV-GFP 및 1 mL의 EF1-GFP 상청액을 사용하였다). (N=2).
도 45. 악성 형질 세포에서의 리간드 수용체 상호 작용. APRIL 리간드는 TAC1 또는 BCMA와 결합한다. BAFF 리간드는 TAC1, BCMA 또는 BAFF-R와 결합한다.
본 개시내용은 키메라 항원 수용체 (CAR)의 조성물, 이의 제조 및 사용 방법을 제공한다.
키메라 항원 수용체 (CAR) 폴리펩타이드는 신호 펩타이드, 항원 인식 도메인, 힌지 영역, 막통과 도메인, 적어도 하나의 보조 자극 도메인 및 신호전달 도메인을 포함한다.
제1세대 CAR은 CD3z를 세포내 신호전달 도메인으로서 포함하는 반면, 제2세대 CAR은 다양한 단백질에서 유래된 적어도 하나의 단일 보조 자극 도메인을 포함한다. 보조 자극 도메인의 예는 CD28, CD2, 4-1BB (CD137, "4-BB"라고도 지칭됨) 및 OX-40 (CD124)을 포함하나 이에 한정되지 않는다. 제3세대 CAR은 두 가지 보조 자극 도메인, 예를 들어, 제한없이 CD28, 4-1BB, CD134 (OX-40), CD2 및/또는 CD137 (4-1BB)을 포함한다.
본원에 사용된 용어 "펩타이드", "폴리펩타이드" 및 "단백질"은 상호교환적으로 사용되며, 펩타이드 결합에 의해 공유 연결된 아미노산 잔기를 갖는 화합물을 지칭한다. 단백질 또는 펩타이드는 적어도 2 개의 아미노산을 포함해야 하며, 단백질 또는 펩타이드의 서열을 포함할 수 있는 아미노산의 최대 수에 제한을 두지 않는다. 폴리펩타이드는 펩타이드 결합에 의해 서로 결합된 2개 이상의 아미노산을 갖는 임의의 펩타이드 또는 단백질을 포함한다. 본원에 사용된 상기 용어는, 예를 들어 통상적으로 당 업계에서 펩타이드, 올리고펩타이드 및 올리고머로도 지칭되는 단쇄뿐만 아니라 일반적으로 당 업계에서 단백질로 지칭되며 많은 종류가 존재하는 장쇄도 지칭한다. "폴리펩타이드"는 예를 들어 생물학적 활성 단편, 실질적으로 상동 폴리펩타이드, 올리고펩타이드, 동종이합체, 이질이합체, 폴리펩타이드의 변이체, 변형된 폴리펩타이드, 유도체, 유사체, 융합단백질 등을 포함한다. 폴리펩타이드는 천연 펩타이드, 재조합 펩타이드, 합성 펩타이드, 또는 이들의 조합물을 포함한다.
하나의 "신호 펩타이드"는 하나의 펩타이드 서열을 포함하는 데 이는 펩타이드 및 임의의 세포 내의 부착된 폴리펩타이드의, 예를 들어 특정 세포소기관 (예를 들어, 소포체) 및/또는 세포 표면으로의 수송과 배치를 지시한다.
신호 펩타이드는 임의의 분비된 또는 막통과 단백질의 펩타이드인데 그것은 본 개시내용의 폴리펩타이드의 세포막 및 세포 표면으로의 수송을 지시하고, 본 개시내용의 폴리펩타이드의 정확한 배치를 제공한다. 특히, 본 개시내용의 신호 펩타이드는 본 개시내용의 폴리펩타이드를 세포막으로 향하게 하며, 여기서 폴리펩타이드의 세포외 부분은 세포 표면 상에 드러나 있고, 막통과 부분은 원형질 막에 걸쳐 있고, 활성 도메인은 세포질 부분 안에, 또는 세포의 내부에 있다.
하나의 실시양태에서, 신호 펩타이드는 소포체 (ER)를 통과한 후에 절단되는, 즉 절단 가능한 신호 펩타이드이다. 한 실시양태에서, 신호 펩타이드는 유형 I, II, III 또는 IV의 인간 단백질이다. 한 실시양태에서, 신호 펩타이드는 면역글로불린 중쇄 신호 펩타이드를 포함한다.
"항원 인식 도메인"은 표적의 항원, 수용체, 펩타이드 리간드 또는 단백질 리간드에 대해 선택적이거나 또는 표적하는 폴리펩타이드 또는 표적의 폴리펩타이드를 포함한다.
항원 인식 도메인은 리간드 결합 및/또는 신호 전달과 연관된 임의의 매우 다양한 세포외 도메인 또는 분비된 단백질로부터 얻을 수 있다. 항원 인식 도메인은 Ig 경쇄 일부와 연결된 Ig 중쇄 부분을 포함할 수 있고 표적 항원에 특이적으로 결합하는 단일 사슬 단편 변이체 (scFv)를 구성할 수 있다. 항체는 단클론항체 또는 다클론항체이거나 또는 표적 항원에 특이적으로 결합하는 임의의 유형일 수 있다. 또 다른 실시 양태에서, 항원 인식 도메인은 수용체 또는 리간드일 수 있다. 특정 실시 양태에서, 표적 항원은 특정 질환 상태에 특이적이고 질환 상태는 화합물 CAR 아키텍처에 존재하는 키메라 수용체 구축물 중 적어도 하나에 의해 인지될 수 있는 세포 표면 항원을 갖는 한 임의의 종류일 수 있다. 특정 실시 양태에서, 키메라 수용체는 특정 단클론 항체 또는 다클론 항체가 존재하거나 생성될 수 있는 임의의 암을 위한 것일 수 있다. 특히, 신경모세포종, 소세포폐암, 흑색종, 난소암, 신장세포암종, 대장암, 호지킨림프종 및 소아 급성 림프모구성백혈병과 같은 암은 키메라 수용체에 특이적인 항원을 갖는다.
표적 특이적 항원 인식 도메인은 바람직하게도, 표적 항원에 대한 항체, 또는 표적 항원에 결합하는 펩타이드, 또는 표적 항원에 결합하는 항체에 결합하는 펩타이드 또는 단백질, 또는 표적 상 수용체에 결합하는 펩타이드 또는 단백질 리간드 (성장 인자, 사이토카인 또는 호르몬을 포함하나 이에 한정되지는 않음)로부터 유래된 항원 결합 도메인을 포함하거나, 또는 표적상의 펩타이드 또는 단백질 리간드에 결합하는 수용체 (성장 인자 수용체, 사이토카인 수용체 또는 호르몬 수용체를 포함하나 이에 한정되지는 않음)로부터 유래된 도메인을 포함한다.
하나의 실시양태에서, 항원 인식 도메인은 표적에 대해 (선택적으로) 지시된 단클론항체 또는 다클론항체의 결합 부분 또는 가변 영역을 포함한다.
또 다른 실시 양태에서, 항원 인식 도메인은 낙타류 단일 도메인 항체 또는 이의 일부를 포함한다. 하나의 실시 양태에서, 낙타류 단일 도메인 항체는 낙타류에서 발견되는 중쇄 항체 또는 VHH 항체를 포함한다. 낙타 동물 (예: 낙타, 단봉낙타, 라마 및 알파카)의 VHH 항체는 낙타류 단일 사슬 항체의 가변 단편을 말하며 (Nguyen et al, 2001; Muyldermans, 2001 참조), 또한 낙타류의 단리된 VHH 항체, 낙타류의 재조합 VHH 항체, 또는 낙타류의 합성 VHH 항체를 포함한다.
또 다른 실시 양태에서, 항원 인식 도메인은 그들의 동족 수용체와 결합하는 리간드를 포함한다. 예로서, APRIL은 TAC1 수용체 또는 BCMA 수용체에 결합하는 리간드이다. 본원에 개시된 발명에 따르면, 항원 인식 도메인은 APRIL 또는 이의 단편을 포함한다. 추가의 예로서, BAFF는 BAFF-R 수용체 또는 BCMA 수용체에 결합하는 리간드이다. 본원에 개시된 발명에 따르면, 항원 인식 도메인은 BAFF 또는 이의 단편을 포함한다. 또 다른 실시 양태에서, 항원 인식 도메인은 인간화된다.
항원 인식 도메인은 그의 서열 내에 약간의 가변성을 포함할 수 있고, 본원에 개시된 표적에 대해 여전히 선택적인 것으로 이해된다. 따라서, 항원 인식 도메인의 폴리펩타이드는 본원에 개시된 항원 인식 도메인 폴리펩타이드와 적어도 95%, 적어도 90%, 적어도 80%, 또는 적어도 70% 동일할 수 있으며, 여기에 설명된 표적에 대해 여전히 선택적이고, 본 개시내용의 범위 내에 있다는 것이 고려된다.
표적은 인터루킨 6 수용체, NY-ESO-1, 알파 태아단백 (AFP), 글리피칸-3 (GPC3), BCMA, BAFF-R, TACI, LeY, CD5, CD13, CD14, CD15 CD19, CD20, CD22, CD33, CD41, CD61, CD64, CD68, CD117, CD123, CD138, CD267, CD269, CD38, Flt3 수용체, CS1, CD45, ROR1, PSMA, MAGE A3, 당지질, 글리피칸 3, F77, GD-2, WT1, CEA, HER-2/neu, MAGE-3, MAGE-4, MAGE-5, MAGE- 6, 알파태아단백, CA 19-9, CA 72-4, NY-ESO, FAP, ErbB, c-Met, MART-1, CD30, EGFRvIII, 면역 글로블린 카파 및 람다, CD38, CD52, CD3, CD4, CD8, CD5, CD7, CD2, 및 CD138을 포함한다.
또 다른 실시 양태에서, 표적은 인터루킨 6 수용체, NY-ESO-1, 알파태아단백 (AFP), 글리피칸-3 (GPC3), BCMA, BAFF-R, TACI, LeY, CD5, CD13, CD14, CD15 CD19, CD20, CD22, CD33, CD41, CD61, CD64, CD68, CD117, CD123, CD138, CD267, CD269, CD38, Flt3 수용체, CS1, CD45, TACI, ROR1, PSMA, MAGE A3, 당지질, 글리피칸 3, F77, GD-2, WT1, CEA, HER-2/neu, MAGE-3, MAGE-4, MAGE-5, MAGE- 6, 알파태아단백, CA 19-9, CA 72-4, NY-ESO, FAP, ErbB, c-Met, MART-1, CD30, EGFRvIII, 면역 글로블린 카파 및 람다, CD38, CD52, CD3, CD4, CD8, CD5, CD7, CD2, 및 CD138의 임의의 부분을 포함한다.
하나의 실시 양태에서, 표적은 인터루킨 6 수용체, NY-ESO-1, 알파태아단백 (AFP), 글리피칸-3 (GPC3), BCMA, BAFF-R, TACI, LeY, CD5, CD13, CD14, CD15 CD19, CD20, CD22, CD33, CD41, CD61, CD64, CD68, CD117, CD123, CD138, CD267, CD269, CD38, Flt3 수용체, CS1, CD45, TACI, ROR1, PSMA, MAGE A3, 당지질, 글리피칸 3, F77, GD-2, WT1, CEA, HER-2/neu, MAGE-3, MAGE-4, MAGE-5, MAGE- 6, 알파태아단백, CA 19-9, CA 72-4, NY-ESO, FAP, ErbB, c-Met, MART-1, CD30, EGFRvIII, 면역 글로블린 카파 및 람다, CD38, CD52, CD3, CD4, CD8, CD5, CD7, CD2, 및 CD138 폴리펩타이드의 표면 노출된 부분을 포함한다.
또 다른 실시 양태에서, 표적 항원은 인간 유두종 바이러스 (HPV) 또는 EBV (엡스타인-바 바이러스) 항원으로부터의 E6 및 E7과 같은 바이러스 또는 곰팡이 항원; 이의 일부; 또는 이의 표면 노출된 영역을 포함한다.
하나의 실시 양태에서, TACI 항원 인식 도메인은 SEQ ID NO. 24를 포함한다.
하나의 실시 양태에서, BCMA 항원 인식 도메인은 SEQ ID NO. 25를 포함한다.
하나의 실시 양태에서, CS1 항원 인식 도메인은 SEQ ID NO. 26를 포함한다.
하나의 실시 양태에서, BAFF-R 항원 인식 도메인은 SEQ ID NO. 27를 포함한다.
하나의 실시 양태에서, CD33 항원 인식 도메인은 SEQ ID NO. 28를 포함한다.
하나의 실시 양태에서, CD123 항원 인식 도메인은 SEQ ID NO. 29를 포함한다.
하나의 실시 양태에서, CD19 항원 인식 도메인은 SEQ ID NO. 30를 포함한다.
하나의 실시 양태에서, CD20 항원 인식 도메인은 SEQ ID NO. 31를 포함한다. 또 다른 하나의 실시 양태에서, CD20 항원 인식 도메인은 SEQ ID NO. 32를 포함한다.
하나의 실시 양태에서, CD22 항원 인식 도메인은 SEQ ID NO. 33를 포함한다.
하나의 실시 양태에서, CD45 항원 인식 도메인은 SEQ ID NO. 34를 포함한다.
힌지 영역은 예를 들어, 키메라 항원 수용체 그리고 적어도 하나의 보조 자극 도메인 및 신호전달 도메인을 포함하나 이에 한정되지는 않는 사이에 위치된 서열이다. 힌지 서열은 예를 들어 인간 또는 그 일부를 포함하는 임의의 속으로부터의 임의의 적합한 서열로부터 포함하여 얻어질 수 있다. 이러한 힌지 영역은 당 업계에 공지되어있다. 하나의 실시양태에서, 힌지 영역은 CD-8 알파, CD28, 4-1BB, OX40, CD3-제타, T 세포 수용체 α 또는 β 사슬, CD3 제타 사슬, CD28, CD3ε, CD45, CD4, CD5, CD8, CD8a, CD9, CD16, CD22, CD33, CD37, CD64, CD80, CD86, CD134, CD137, ICOS, CD154, 이들의 기능적 유도체 그리고 이들의 조합을 포함하는 인간 단백질의 힌지 영역을 포함한다.
하나의 실시 양태에서 힌지 영역은 CD8 힌지 영역을 포함한다.
일부 실시 양태에서, 힌지 영역은, 이에 제한되는 것은 아니지만, 면역글로불린 (예를 들어, IgG1, IgG2, IgG3, IgG4 및 IgD)으로부터 선택되는 하나를 포함한다.
막통과 도메인은 세포막에 걸치는 소수성 폴리펩타이드를 포함한다. 특히, 막통과 도메인은 세포막의 한면 (세포외)에서 세포막의 다른면 (세포내 또는 세포질)을 통하여 걸쳐있다.
막통과 도메인은 알파 나선 또는 베타 배럴, 또는 이들의 조합의 형태일 수 있다. 막통과 도메인은 다수의 막통과 절편, 각 알파-나선형, 베타 시트, 또는 이들의 조합을 갖는 다원(polytopic) 단백질을 포함할 수 있다.
하나의 실시 양태에서, CAR에서의 도메인 중 하나와 자연적으로 회합되는 막통과 도메인이 사용된다. 또 다른 실시양태에서, 막통과 도메인은 아미노산 치환에 의해 선택되거나 변형되어, 동일한 또는 상이한 표면 막 단백질의 막통과 도메인에 이러한 도메인이 결합하는 것을 막고 수용체 복합체의 다른 구성원과의 상호 작용을 최소화한다.
예를 들어, 막통과 도메인은 T-세포 수용체 α 또는 β 쇄, CD3 제타 쇄, CD28, CD3ε, CD45, CD4, CD5, CD7, CD8, CD9, CD16, CD22, CD33, CD37, CD64, CD80, CD86, CD68, CD134, CD137, ICOS, CD41, CD154, 이들의 기능적 유도체 및 이들의 조합물의 막통과 도메인을 포함한다.
하나의 실시 양태에서, 막통과 도메인은 도메인의 아미노산 잔기의 25 % 초과, 50 % 초과 또는 75 % 초과가 류신 및 발린과 같은 소수성 잔기가 되도록 인위적으로 설계된다. 하나의 실시 양태에서, 페닐알라닌, 트립토판 및 발린의 삼중체는 합성 막통과 도메인의 각 말단에서 발견된다.
하나의 실시 양태에서, 막통과 도메인은 CD8 막통과 도메인이다. 또 다른 실시 양태에서, 막통과 도메인은 CD28 막통과 도메인이다. 이러한 막통과 도메인은 당 업계에 공지되어있다.
신호전달 도메인 및 보조 자극 도메인은 면역세포 신호전달 경로의 적어도 일부 면을 자극 또는 활성화시키기 위한 면역세포의 활성화를 제공하는 폴리펩타이드를 포함한다.
하나의 실시양태에서, 신호전달 도메인은 CD3 제타, 공통 FcR 감마 (FCER1G), Fc감마RIIIa, FcR베타 (Fc 엡실론 립), CD3감마, CD3델타, CD3 엡실론, CD79a, CD79b, DNAX- 활성화 단백질 10 (DAP10), DNAX- 활성화 단백질 12 (DAP12), 이들의 활성 단편, 이들의 기능적 유도체 및 이들의 조합물의 기능적 신호전달 도메인의 폴리펩타이드를 포함한다. 이러한 신호전달 도메인은 당 업계에 공지되어있다.
하나의 실시 양태에서, CAR 폴리펩타이드는 하나 이상의 보조 자극 도메인을 추가로 포함한다. 하나의 실시 양태에서, 보조 자극 도메인은 OX40; CD27; CD28; CD30; CD40; PD-1; CD2; CD7; CD258; 자연살상 그룹 2 멤버 C (NKG2C); 자연살상 그룹 2 멤버 (NKG2D); B7-H3; CD83, ICAM-1, LFA-1 (CD11a/CD18), ICOS 및 4-1BB (CD137) 중 적어도 하나에 결합하는 리간드, CDS; ICAM-1; LFA-1 (CD1a/CD18); CD40; CD27; CD7; B7-H3; NKG2C; PD-1; ICOS; 이들의 활성 단편, 이들의 기능 유도체, 및 이들의 조합을 포함하는 단백질로부터 유래한 기능 신호전달 도메인이다.
본원에 사용된 바와 같이, 적어도 하나의 보조 자극 도메인 및 신호전달 도메인은 세포내 도메인으로서 총체적으로 지칭될 수 있다. 본원에 사용된 바와 같이, 힌지 영역과 항원 인식은 세포외 도메인으로서 총체적으로 지칭될 수 있다.
본 개시내용은 상기 기술된 키메라 항원 수용체 폴리펩타이드를 코딩하는 폴리뉴클레오타이드를 추가로 제공한다.
본원에 사용된 바와 같이 용어 "폴리뉴클레오타이드"는 뉴클레오타이드의 사슬로서 정의된다. 폴리뉴클레오타이드는 DNA 및 RNA를 포함한다. 더욱이, 핵산은 뉴클레오타이드의 중합체이다. 따라서, 본원에 사용된 핵산 및 폴리뉴클레오타이드는 상호 교환 가능하다. 당업계의 통상의 기술자는 핵산이 폴리뉴클레오타이드고 이것이 단량체 "뉴클레오타이드"로 가수 분해될 수 있다는 일반적인 지식을 갖는다. 단량체 뉴클레오타이드는 뉴클레오사이드로 가수 분해될 수 있다. 본원에 사용된 바와 같이 폴리뉴클레오타이드는 통상적인 클로닝 기술 및 중합 효소 연쇄 반응 (PCR) 등을 사용하는 재조합 수단들, 즉 재조합 라이브러리 또는 세포 유전체로부터의 핵산 서열 클로닝을 포함하나 이에 제한되는 않는, 당업계에서 이용 가능한 임의의 수단, 및 합성 수단에 의해 획득되는 모든 핵산 서열을 포함하나 그것에 국한되는 것은 아니다.
CAR을 코딩하는 폴리뉴클레오타이드는 임의의 통상적인 방법에 의해 특정 CAR의 아미노산 서열로부터 용이하게 제조된다. 아미노산 서열을 코딩하는 염기 서열은 전술한 각 도메인의 아미노산 서열에 대한 전술된 NCBI RefSeq ID 또는 GenBenk의 수탁번호로부터 얻을 수 있으며, 본 개시내용의 핵산은 표준 분자 생물학적 및/또는 화학적 절차를 이용하여 제조될 수 있다. 예를 들어, 염기 서열에 기초하여 폴리뉴클레오타이드를 합성할 수 있고, 중합 효소 연쇄 반응 (PCR)을 이용하여 cDNA 라이브러리로부터 얻어지는 DNA 단편을 조합하는 것에 의해 본 개시내용의 폴리뉴클레오타이드를 제조할 수 있다.
하나의 실시 양태에서, 본원에 개시된 폴리뉴클레오타이드는 유전자, 또는 발현 또는 클로닝 카세트의 일부이다.
상기 기재된 폴리뉴클레오타이드는 벡터로 클로닝될 수 있다. "벡터"는 단리된 폴리뉴클레오타이드를 포함하고 단리된 폴리뉴클레오타이드를 세포의 내부로 전달하는데 사용될 수 있는 물질의 조성물이다. 선형 폴리뉴클레오타이드, 이온 또는 양친매성 화합물과 회합된 폴리뉴클레오타이드, 플라스미드, 파지미드, 코스미드 및 바이러스를 포함하는 많은 벡터가 당 업계에 공지되어 있지만, 이에 한정되는 것은 아니다. 바이러스는 파지, 파지 유도체를 포함한다. 따라서, 용어 "벡터"는 자율적으로 복제하는 플라스미드 또는 바이러스를 포함한다. 이 용어는 또한 예를 들어, 폴리라이신 화합물, 리포솜 등과 같은, 세포로의 핵산의 전달을 용이하게 하는 비플라스미드 및 비바이러스 화합물을 포함하는 것으로 해석되어야 한다. 바이러스 벡터의 예는 아데노바이러스 벡터, 아데노-연관 바이러스 벡터, 레트로바이러스 벡터, 렌티바이러스 벡터 등을 포함하나 이에 한정되지는 않는다. 하나의 실시 양태에서, 벡터는 클로닝 벡터, 발현 벡터, 복제 벡터, 프로브 생성 벡터, 통합 벡터, 그리고 시퀀싱 벡터를 포함한다.
하나의 실시 양태에서, 벡터는 바이러스 벡터다. 하나의 실시 양태에서, 바이러스 벡터는 레트로바이러스 벡터 또는 렌티바이러스 벡터다. 하나의 실시 양태에서, 조작된 세포는 바이러스에 의해 형질도입되어 폴리뉴클레오타이드 서열을 발현한다.
많은 바이러스 기반 시스템이 포유동물 세포로의 유전자 전달을 위해 개발되었다. 예를 들어, 레트로바이러스는 유전자 전달 시스템을 위한 편리한 플랫폼을 제공한다. 선택된 유전자는 당업계에 공지된 기술을 사용하여 벡터에 삽입되고 레트로바이러스 입자 내에 패키징될 수 있다. 이어서, 재조합 바이러스는 단리되고 생체내 또는 생체외에서 환자 세포로 전달될 수 있다. 다수의 레트로바이러스 시스템이 당 업계에 공지되어 있다. 일부 실시 양태에서, 아데노바이러스 벡터가 사용된다. 다수의 아데노바이러스 벡터가 당 업계에 공지되어 있다. 하나의 실시 양태에서, 렌티바이러스 벡터가 사용된다.
바이러스 벡터 기술은 당업계에 잘 공지되어 있으며, 예를 들어 Sambrook 등 (2001, Molecular Cloning: A Laboratory Manual, Cold Spring Harbor Laboratory, New York), 그리고 기타 바이러스학 및 분자 생물학 매뉴얼에 기술되어 있다. 벡터로서 유용한 바이러스는 레트로바이러스, 아데노바이러스, 아데노-연관 바이러스, 헤르페스바이러스 및 렌티바이러스를 포함하나 이에 한정되지는 않는다. 일반적으로, 적합한 벡터는 적어도 하나의 유기체에서 기능적인 복제 기점, 프로모터 서열, 편리한 제한 엔도뉴클레아제 부위 및 하나 이상의 선택 마커를 포함한다 (예를 들어, WO 01/96584; WO 01/29058; 및 U.S. Pat. No. 6,326,193).
렌티바이러스 벡터는 유전자를 인간 T 세포에 높은 효율로 전달하는 능력으로 잘 알려져 있지만 벡터-코딩된 유전자의 발현은 그들의 발현을 유도하는 내부 프로모터에 의존한다. 증가된 CAR 바디 사이즈가 동일한 수준의 발현을 보장하지 못하기 때문에 강력한 프로모터는 추가적인 보조 자극 도메인 또는 증식성 사이토카인을 코딩하는 유전자를 보유하는 제3세대 또는 4세대 CAR에 특히 중요하다. 상이한 강도와 세포 유형 특이성을 갖는 광범위한 프로모터가 존재한다. CAR T 세포를 이용하는 유전자 요법은 충분한 CAR 바디를 발현하고 장기간에 걸쳐 발현을 유지하는 T 세포의 능력에 의존한다. EF-1α 프로모터가 CAR 발현을 위해 통상적으로 선택되었다.
본 발명은 T 세포 또는 NK 세포에서의 높은 수준의 유전자 발현을 위한 강력한 프로모터를 함유하는 발현 벡터에 관한 것이다. 추가의 실시 양태에서, 발명자는 T 세포 또는 NK 세포에서 CAR의 고수준 발현에 유용한 강력한 프로모터를 개시한다. 특정 실시 양태에서, 강한 프로모터는 T 세포 또는 NK 세포에서 높은 수준의 발현을 얻고 장시간 동안 발현을 유지하기 위해 발현 벡터에 선택적으로 도입되는 SFFV 프로모터에 관한 것이다. 발현된 유전자는 면역 요법에 사용되는 CAR, T 세포 보조 자극 인자 및 사이토 카인을 선호한다.
적합한 프로모터의 한 예는 극초기 사이토메갈로바이러스 (CMV) 프로모터 서열이다. 이 프로모터 서열은 그에 작동 가능하게 연결된 임의의 폴리뉴클레오타이드 서열의 높은 수준의 발현을 유도할 수 있는 강력한 구성적 프로모터 서열이다. 적합한 프로모터의 다른 예는 신장 성장 인자-1a (EF-1a)이다. 그러나, 원숭이바이러스 40 (SV40) 초기 프로모터, 마우스 유방 종양 바이러스 (MMTV), 인간 면역 결핍 바이러스 (HIV) 긴 말단 반복부 (LTR) 프로모터, MoMuLV 프로모터, 조류백혈병 바이러스 프로모터, 엡스타인-바 바이러스 극초기 프로모터, 라우스 육종 바이러스 프로모터뿐만 아니라, 예컨대 비제한적으로 액틴 프로모터, 미오신 프로모터, 혈색소 프로모터, 및 크레아틴 키나아제 프로모터인 인간 유전자 프로모터를 포함하나 이에 한정되지는 않는 다른 구성적 프로모터 서열이 또한 사용될 수 있다. 또한, 본 개시내용은 구성적 프로모터의 사용으로 제한되지 않아야 하며, 유도성 프로모터도 본 개시내용의 일부로서 고려된다. 유도성 프로모터의 사용은 발현이 요구될 때 작동 가능하게 연결된 폴리뉴클레오타이드 서열의 발현을 작동시킬 수 있거나 발현이 요구되지 않을 때 발현을 끌 수 있는 분자 스위치를 제공한다. 유도성 프로모터의 예로는 메탈로티오네인 프로모터, 글루코코르티코이드 프로모터, 프로게스테론 프로모터 및 테트라사이클린 프로모터를 포함하나, 이에 한정되는 것은 아니다.
키메라 항원 수용체 폴리뉴클레오타이드의 발현은 예를 들어, 적어도 하나의 SFFV (비장-포커스 형성 바이러스) (예를 들어, SEQ ID NO. 23) 또는 인간 신장 인자 11α (EF) 프로모터, CAG (CMV 인핸서를 갖는 닭 베타-액틴 프로모터) 프로모터, 인간 신장 인자 1α (EF) 프로모터를 포함하나 이에 한정되지는 않는 발현 벡터를 사용하여 달성될 수 있다. 이용된 덜 강력한/저 발현 프로모터의 예는 원숭이바이러스40 (SV40) 초기 프로모터, 사이토메갈로바이러스 (CMV) 극초기 프로모터, 유비퀴틴 C (UBC) 프로모터 및 포스포글리세레이트키나제 1 (PGK) 프로모터 또는 이들의 일부를 포함하나 이에 한정되지는 않는다. 키메라 항원 수용체의 유도성 발현은 예를 들어, TRE3GV (모든 세대 및 바람직하게는 제 3 세대를 비롯한 Tet-반응 요소), 유도성 프로모터 (Clontech Laboratories, Mountain View, CA), 또는 이들의 일부 또는 조합을 포함하나 이에 한정되지는 않는 테트라사이클린 반응성 프로모터를 사용하여 달성될 수 있다.
바람직한 실시 양태에서, 프로모터는 SFFV 프로모터 또는 이의 유도체이다. 본 개시내용에 준하여 SFFV 프로모터가 본 개시내용에 따른 형질 도입된 세포에서 더 강한 발현 및 더 큰 지속성을 제공한다는 사실이 예상치 못하게 밝혀졌다.
"발현 벡터"는 발현된 뉴클레오타이드 서열에 작동 가능하게 연결된 발현 조절 서열을 포함하는 재조합 폴리뉴클레오타이드를 포함하는 벡터를 말한다. 발현 벡터는 발현을 위한 충분한 시스-작용 요소를 포함한다; 발현을 위한 다른 요소는 숙주 세포에 의해 또는 시험관내 발현 시스템 내에서 공급될 수 있다. 발현 벡터는 재조합 폴리뉴클레오타이드를 포함하는 코스미드, 플라스미드 (예를 들어, 네이키드 또는 리포솜 내에 함유됨) 및 바이러스 (예를 들어, 렌티바이러스, 레트로바이러스, 아데노바이러스 및 아데노-연관 바이러스)와 같은 당 업계에 공지된 모든 것들을 포함한다. 발현 벡터는 바이시스트론 (bicistronic) 또는 멀티시스트론 (multicistronic) 발현 벡터일 수 있다. 바이시스트론 (bicistronic) 또는 멀티시스트론 (multicistronic) 발현 벡터는 (1) 각각의 오픈 리딩 프레임에 융합된 다수의 프로모터; (2) 유전자들 사이에 스플라이싱 신호 삽입; 발현이 단일 프로모터에 의해 유도되는 유전자의 융합; (3) 유전자들 사이에 단백질분해 절단 부위의 삽입 (자가 절단 펩타이드); 및 (iv) 유전자들 사이에 내부 리보솜 진입 부위 (IRES)의 삽입.
하나의 실시 양태에서, 본 개시내용은 적어도 하나의 키메라 항원 수용체 폴리펩타이드 또는 폴리뉴클레오타이드를 갖는 조작된 세포를 제공한다.
"조작된 세포"는 유전자, DNA 또는 RNA 서열, 또는 단백질 또는 폴리펩타이드의 첨가 또는 변형에 의해 변형, 형질전환 또는 조작된 임의의 유기체의 임의의 세포를 의미한다. 본 개시내용의 단리된 세포, 숙주 세포 및 유전적으로 조작된 세포는 키메라 항원 수용체 또는 키메라 항원 수용체 복합체를 코딩하는 DNA 또는 RNA 서열을 함유하는 NK세포 및 T세포와 같은 단리된 면역 세포를 포함하고, 세포 표면에서 키메라 수용체를 발현한다. 단리된 숙주 세포 및 조작된 세포는 예를 들어, NK 세포 활성 또는 T 림프구 활성의 증진, 암 치료, 및 감염질환 치료를 위해 사용될 수 있다.
하나의 실시 양태에서, 조작된 세포는 면역조절 세포를 포함한다. 면역조절 세포에는 CD4 T세포 (헬퍼T세포), CD8T세포 (세포독성T세포, CTL), 및 기억T세포 또는 기억 줄기세포T세포와 같은 T세포가 포함된다. 또 다른 실시 양태에서, T세포는 자연 살상 T세포 (NK T세포)를 포함한다.
하나의 실시 양태에서, 조작된 세포는 자연 살상 세포를 포함한다. 자연 살상 세포는 당 업계에 잘 알려져 있다. 하나의 실시 양태에서, 자연 살상 세포는 NK-92 세포와 같은 세포주를 포함한다. NK 세포주의 추가 예는 NKG, YT, NK-YS, HANK-1, YTS 세포 및 NKL 세포를 포함한다.
NK 세포는 GvHD의 위험없이 항종양 효과를 중개하고 T 세포에 비해 수명이 짧다. 그러므로, 암세포를 파괴한 직후 NK세포는 소진되고, 이는 변형된 세포를 제거할 수 있는 CAR 구축물 상의 유도성 자살 유전자에 대한 필요를 감소시킬 것이다.
본 개시내용에 따르면, 놀랍게도, NK 세포가 본원에 개시된 키메라 항원 수용체 폴리펩타이드를 함유하고 발현하도록 조작될 용이하게 이용가능한 세포를 제공한다는 것이 놀랍게 밝혀졌다.
동종이계 또는 자가 NK 세포는 빠른 면역 반응을 유도하지만 그들의 제한된 수명으로 인해 혈액순환으로부터 상대적으로 빠르게 사라진다. 따라서, 본 출원인들은 놀랍게도 CAR 세포 기반 요법을 사용하면 부작용 지속의 우려가 감소된다는 것을 발견했다.
본 개시내용은 cCAR을 생성하는 방법을 포함한다. 일부 실시 양태에서, cCAR은 T 세포를 사용하여 생성된다. 다른 실시 양태에서, cCAR은 질환 또는 암을 가진 임의의 포유동물에게 "기성품(off-the-shelf)"으로 투여되도록 말초 혈액 또는 제대혈 및 NK-92 세포로부터 단리된 1차 NK 세포를 사용한다.
본 발명의 한 양태에 따르면, NK 세포는 본 발명에 따라 팽창되고 CAR 폴리뉴클레오타이드로 형질 감염될 수 있다. NK 세포는 제대혈, 말초 혈액, iPS 세포 및 배아 줄기 세포에서 유래될 수 있다. 본 발명의 한 양태에 따르면, NK-92 세포는 팽창되고 CAR로 형질 감염될 수 있다. NK-92는 자연 살상 세포 (NK 세포)의 특색과 특징을 가진 지속적으로 성장하는 세포주이다 (Arai, Meagher et al., 2008). NK-92 세포주는 IL-2 의존적이며 안전하고 (Arai, Meagher 등, 2008) 실현가능한 것으로 입증되었다. CAR 발현하는 NK-92 세포는 무혈청-배지에서 피더 세포와 공동 배양하거나 공동 배양하지 않는 것으로 팽창될 수 있다. 관심 CAR을 운반하는 NK-92의 순수한 집단은 분류에 의해 얻을 수 있다.
하나의 실시 양태에서, 조작된 세포는 공여자로부터 얻은, MHC 인식에 관여하는 TCR (T 세포 수용체)의 성분을 불활성화하도록 변형된, 동종이계 T 세포를 포함한다. 결과적으로, TCR 결핍 T 세포는 이식편 대 숙주 질환 (GVHD)을 유발하지 않을 것이다.
일부 실시 양태에서, 조작된 세포는 세포 표면 항원의 발현을 방지하도록 변형될 수 있다. 예를 들어, 조작된 세포는 천연 CD45 유전자를 제거하도록 유전적으로 변형되어 이들의 발현 및 세포 표면 디스플레이를 방지할 수 있다.
일부 실시 양태에서, 조작된 세포는 유도성 자살 유전자 ("안전 스위치") 또는 안전 스위치들의 조합을 포함하며, 이는 비제한적으로 레트로바이러스 벡터, 렌티바이러스 벡터, 아데노바이러스 벡터 또는 플라스미드와 같은 벡터 상에서 조립될 수 있다. "안전 스위치"의 도입은 안전 프로필을 크게 증가시키고 화합물 CAR의 온-표적(on-target) 또는 오프-종양(off-tumor) 독성을 제한한다. "안전 스위치"는 비제한적으로 카스파제 9 유전자, 티미딘키나아제, 시토신데아미나제 (CD) 또는 시토크롬 P450과 같은 유도성 자살 유전자일 수 있다. 원하지 않는 변형된 T 세포를 제거하기 위한 다른 안전 스위치는 T 세포에서의 CD20 또는 CD19의 발현 또는 말단절단된 표피 성장 인자 수용체를 포함한다. 본 발명에서는 가능한 모든 안전 스위치가 고려되고 실시된다.
일부 실시 양태에서, 자살 유전자는 조작된 세포 유전체 내로 통합된다.
하나의 실시 양태에서, 본 개시내용은 CD45 키메라 항원 수용체 폴리뉴클레오타이드를 갖는 조작된 세포를 제공한다. 하나의 실시 양태에서, CD45 CAR 폴리펩타이드는 SEQ ID NO.13 및 상응하는 폴리뉴클레오타이드 서열 SEQ ID NO. 14를 포함한다. 또 다른 실시 양태에서, CD45 CAR 폴리펩타이드는 SEQ ID NO. 15 및 상응하는 폴리뉴클레오타이드 서열 SEQ ID NO. 16을 포함한다. 또 다른 실시 양태에서, CD45 CAR 폴리펩타이드는 SEQ ID NO. 17 및 상응하는 폴리뉴클레오타이드 서열 SEQ ID NO. 18을 포함한다.
다중 CAR 단위
본 개시내용은 적어도 2 개의 별개의 CAR 폴리펩타이드를 갖는 조작된 세포를 제공한다.
본원에서 사용된 바와 같이, 화합물 CAR (cCAR) 또는 다중 CAR은 적어도 2개의 별개의 키메라 항원 수용체 폴리펩타이드를 갖는 조작된 세포를 지칭한다. 본원에서 사용된 바와 같이, "별개의 키메라 항원 수용체 폴리펩타이드"는 고유한 항원 인식 도메인, 신호 펩타이드, 힌지 영역, 막통과 도메인, 적어도 하나의 보조 자극 도메인 및 신호전달 도메인을 갖는다. 따라서, 두 개의 고유한 키메라 항원 수용체 폴리펩타이드는 상이한 항원 인식 도메인을 가질 것이다. 신호 펩타이드, 힌지 영역, 막통과 도메인, 적어도 하나의 보조 자극 도메인 및 신호전달 도메인은 2개의 별개의 키메라 항원 수용체 폴리펩타이드 사이에서 동일하거나 상이할 수 있다. 본원에서 사용된 바와 같이, 키메라 항원 수용체 (CAR) 단위는 별개의 키메라 항원 수용체 폴리펩타이드 또는 이를 코딩하는 폴리뉴클레오타이드를 지칭한다.
본원에 사용된 바와 같이, 고유한 항원 인식 도메인은 단일 표적에 대해 특이적이거나 또는 단일 표적을 표적으로 하는 것이거나 또는 표적의 단일 에피토프이다.
일부 실시 양태에서, 화합물 CAR은 동일한 항원을 표적으로 한다. 예를 들어, cCAR은 단일 항원의 상이한 에피토프 또는 부분을 표적으로 한다. 일부 실시 양태에서, 화합물 CAR에 존재하는 각각의 CAR 단위는 질환 상태에 의해 야기되는 동일한 또는 상이한 질환 상태 또는 부작용에 대해 특이적인 상이한 항원을 표적으로 한다.
일부 실시 양태에서, 화합물 CAR은 두 개의 상이한 항원을 표적으로 한다.
상이한 CAR 단위를 지닌 화합물 CAR의 생성은 상당히 어려울 수 있다: (1) CAR-CAR 상호 작용은 해로운 효과가 있을 수 있으며 적절한 CAR 설계가 이 효과를 상쇄하는 키이다; (2) 단일 구축물 내의 화합물 CAR은 발현 카세트의 길이를 증가시킬 수 있고, 이는 바이러스 역가와 단백질 발현 수준의 감소를 일으킬 수 있다; (3) 특히 다중 CAR을 단일 벡터에서 발현시키는 전략을 선택하기 위해 다양한 CAR 바디 요소를 포함하는 적절한 설계가 필요하다; (4) 강력한 프로모터는 CAR의 추가 단위를 지니고 있는 화합물 CAR에 대해 특히 중요하다; (5) CAR 내의 힌지 영역은 각 CAR 단위 사이의 힌지 영역의 상호 작용이 바람직하게 방지되게 설계될 필요가 있다; (6) 세포 내에서 발현하는 CAR의 2 개 이상의 단위는 독성 효과를 일으킬 수 있다 (CAR-CAR 상호 작용). 본 출원인은 이러한 장애물을 극복하기 위해 신규하고 놀라운 CAR 조성물 및 방법을 본원에서 제공한다.
하나의 실시 양태에서, 본 개시내용은 다중 CAR 단위를 갖는 조작된 세포를 제공한다. 이는 단일 조작된 세포가 다수 항원을 표적으로 할 수 있게 허락한다. 다중 CAR 단위를 이용하여 다수 표면 마커 또는 항원을 동시에 표적으로 삼는 것은 내성 클론의 선택을 방지하고 종양의 재발을 줄인다. 다양한 도메인과 활성 부위를 포함하는 각 개별 구성요소 CAR를 이용하는 다중 CAR T세포 면역요법은 아직 어떤 악성 종양에 대해서도 개발되지 않았다.
본 발명의 한 측면에서, cCAR은 다중 CAR 단위를 포함한다. 일부 실시 양태에서, cCAR은 적어도 2개의 CAR 단위를 포함한다. 또 다른 실시 양태에서, cCAR은 적어도 3개의 CAR 단위를 포함한다. 또 다른 실시 양태에서, cCAR은 적어도 4 개의 단위를 포함한다.
하나의 실시 양태에서, 본 개시내용은 각각 상이한 항원 인식 도메인을 갖는, 적어도 2 개의 별개의 키메라 항원 수용체 폴리펩타이드를 갖는 조작된 세포를 제공한다.
바람직한 실시 양태에서, 적어도 2개의 별개의 키메라 항원 수용체 폴리펩타이드를 갖는 조작된 세포는 말초 혈액 또는 제대혈 및 NK-92 세포로부터 단리된 1차 NK 세포이며, 이로 인해 이들은 질환이나 암을 가진 임의의 포유동물에게 "기성품"으로 투여된다.
하나의 실시 양태에서, 조작된 세포는 (i.) 제1항원 인식 도메인, 제1신호 펩타이드, 제1힌지 영역, 제1막통과 도메인, 제1 보조 자극 도메인 및 제1신호전달 도메인을 포함하는 제1키메라 항원 수용체 폴리펩타이드; 및 (ii.) 제2항원 인식 도메인, 제2신호 펩타이드, 제2힌지 영역, 제2막통과 도메인, 제2보조 자극 도메인 및 제2신호전달 도메인을 포함하는 제2키메라 항원 수용체 폴리펩타이드를 포함한다. 제1항원 인식 도메인은 제2항원 인식 도메인과 다르다.
바람직한 실시 양태에서, 각각의 조작된 CAR 단위 폴리뉴클레오타이드는 상동 재조합을 피하기 위해 상이한 뉴클레오타이드 서열을 갖는다.
하나의 실시 양태에서, 제1항원 인식 도메인의 표적은 인터루킨 6 수용체, NY-ESO-1, 알파 태아 단백 (AFP), 글리피칸-3 (GPC3), BAFF-R, BCMA, TACI, LeY, CD5, CD13, CD14, CD15 CD19, CD20, CD22, CD33, CD41, CD61, CD64, CD68, CD117, CD123, CD138, CD267, CD269, CD38, Flt3 수용체, 및 CS1로 이루어진 그룹으로부터 선택되고; 제2인식 도메인의 표적은 인터루킨 6 수용체, NY-ESO-1, 알파 태아 단백 (AFP), 글리피칸-3 (GPC3), BAFF-R, BCMA, TACI, LeY, CD5, CD13, CD14, CD15 CD19, CD20, CD22, CD33, CD41, CD61, CD64, CD68, CD117, CD123, CD138, CD267, CD269, CD38, Flt3 수용체, 및 CS1로 이루어진 그룹으로부터 선택된다.
하나의 실시 양태에서, 조작된 세포는 CD19 항원 인식 도메인을 갖는 제1키메라 항원 수용체 폴리펩타이드 및 CD20 인식 도메인을 갖는 제2키메라 항원 수용체 폴리펩타이드를 포함한다. 하나의 실시 양태에서, 이러한 조작된 세포는 SEQ ID NO. 3의 폴리펩타이드 및 SEQ ID NO. 4의 상응하는 폴리뉴클레오타이드를 포함한다.
하나의 실시 양태에서, 조작된 세포는 CD19 항원 인식 도메인을 갖는 제1키메라 항원 수용체 폴리펩타이드 및 CD22 항원 인식 도메인을 갖는 제2키메라 항원 수용체 폴리펩타이드를 포함한다. 하나의 실시 양태에서, 이러한 조작된 세포는 SEQ ID NO. 5의 폴리펩타이드 및 SEQ ID NO. 6의 상응하는 폴리뉴클레오타이드를 포함한다.
하나의 실시 양태에서, 조작된 세포는 CD19 항원 인식 도메인을 갖는 제1키메라 항원 수용체 폴리펩타이드 및 CD123 항원 인식 도메인을 갖는 제2키메라 항원 수용체 폴리펩타이드를 포함한다. 하나의 실시 양태에서, 이러한 조작된 세포는 SEQ ID NO. 7의 폴리펩타이드 및 SEQ ID NO. 8의 상응하는 폴리뉴클레오타이드를 포함한다.
하나의 실시 양태에서, 조작된 세포는 CD33 항원 인식 도메인을 갖는 제1키메라 항원 수용체 폴리펩타이드 및 CD123 항원 인식 도메인을 갖는 제2키메라 항원 수용체 폴리펩타이드를 포함한다. 하나의 실시 양태에서, 이러한 조작된 세포는 SEQ ID NO. 9의 폴리펩타이드 및 SEQ ID NO. 10의 상응하는 폴리뉴클레오타이드를 포함한다. 또 다른 실시 양태에서, 이 조작된 세포는 SEQ ID NO. 11의 폴리펩타이드 및 SEQ ID NO. 12의 상응하는 폴리뉴클레오타이드를 포함한다.
하나의 실시 양태에서, 조작된 세포는 BAFF-R 항원 인식 도메인을 갖는 제1키메라 항원 수용체 폴리펩타이드 및 CS1 항원 인식 도메인을 갖는 제2키메라 항원 수용체 폴리펩타이드를 포함한다.
하나의 실시 양태에서, 조작된 세포는 CD269 항원 인식 도메인을 갖는 제1키메라 항원 수용체 폴리펩타이드 및 CS1 항원 인식 도메인을 갖는 제2키메라 항원 수용체 폴리펩타이드를 포함한다. 하나의 실시 양태에서, 조작된 세포는 SEQ ID NO. 19의 폴리펩타이드 및 SEQ ID NO. 20의 상응하는 폴리뉴클레오타이드를 포함한다. 하나의 실시 양태에서, 조작된 세포는 SEQ ID NO. 21의 폴리펩타이드 및 SEQ ID NO. 22의 상응하는 폴리뉴클레오타이드를 포함한다.
하나의 실시 양태에서, 조작된 세포는 CD33 항원 인식 도메인을 갖는 제1키메라 항원 수용체 폴리펩타이드 및 CD123 항원 인식 도메인을 갖는 제2키메라 항원 수용체 폴리펩타이드를 포함한다.
하나의 실시 양태에서, 각 CAR 단위는 동일하거나 상이한 힌지 영역을 포함한다. 또 다른 실시 양태에서, 각 CAR 단위는 동일하거나 상이한 막통과 영역을 포함한다. 또 다른 실시 양태에서, 각 CAR 단위는 동일하거나 상이한 세포내 도메인을 포함한다.
하나의 실시 양태에서, 각 CAR 단위는 CD3 제타 쇄 신호전달 도메인을 포함한다.
하나의 실시 양태에서, 각 별개의 CAR 단위는 상호 작용을 피하기 위해 상이한 보조 자극 도메인을 포함한다. 예를 들어, 제1키메라 항원 수용체 폴리펩타이드는 4-BB 보조- 자극 도메인을 포함하고; 제2키메라 항원 수용체 폴리펩타이드는 CD28 보조- 자극 도메인을 포함한다.
또 다른 실시 양태에서, 힌지 영역은 원치 않는 분자내 또는 분자간 상호 작용을 일으킬 수 있는 아미노산을 배제하도록 설계된다. 예를 들어, 힌지 영역은 디술피드 결합의 형성을 방지하기 위해 시스테인 잔기를 배제하거나 최소화하도록 설계될 수 있다. 또 다른 실시 양태에서, 힌지 영역은 원하지 않는 소수성 상호 작용을 방지하기 위해 소수성 잔기를 배제하거나 최소화하도록 설계될 수 있다.
화합물 CAR은 독립적으로 또는 조합하여 살상을 수행할 수 있다. 다중 또는 화합물 CAR은 동일하거나 상이한 힌지 영역, 동일하거나 상이한 막통과, 동일하거나 상이한 보조- 자극 및 동일하거나 상이한 세포내 도메인들을 포함한다. 바람직하게는, 힌지 영역은 상호 작용 부위를 피하도록 선택된다.
본 발명의 화합물 CAR은 T 또는 NK 세포에서 동일하거나 상이한 종양 집단을 표적화할 수 있다. 제1 CAR은 예를 들어, 벌크 종양 집단을 표적으로 할 수 있으며 다음 또는 제2 CAR은 예를 들어, 암 또는 백혈병 줄기 세포를 근절하여 암 재발을 피할 수 있다.
본 발명에 따르면, 놀랍게도 상이한 또는 동일한 종양 집단을 표적으로 하는 T 또는 NK 세포에서의 화합물 CAR은 암세포가 CAR 살상 활성에 저항성이 되도록 하여 암세포 표면으로부터의 표적 항원의 하향 조절을 생성하는 종양 인자를 퇴치한다는 것이 발견되었다. "항원 회피"로 불리는 것이 암세포를 CAR 요법으로부터 "숨겨 지도록" 할 수 있다는 것과 서로 다른 종양 세포가 별개의 표면 항원 발현 프로파일을 보일 수 있게 하는 종양이질성이 또한 놀랍게도 발견되었다.
CAR
폴리펩타이드
및
인핸서를
갖는 조작된 세포
또 다른 실시 양태에서, 본 개시내용은 적어도 하나의 키메라 항원 수용체 폴리펩타이드 및 인핸서를 갖는 조작된 세포를 제공한다.
하나의 실시 양태에서, 본 개시내용은 적어도 2개의 별개의 키메라 항원 수용체 폴리펩타이드 및 인핸서를 갖는 조작된 세포를 제공한다.
본원에 사용된 바와 같이, 인핸서는 키메라 항원 수용체 폴리펩타이드를 갖는 조작된 세포의 활성을 촉진하거나 증진시키는 생물학적 분자를 포함한다. 인핸서에는 사이토카인이 포함된다. 또 다른 실시 양태에서, 인핸서는 IL-2, IL-7, IL-12, IL-15, IL-21, PD-1, PD-L1, CSF1R, CTAL-4, TIM-3 및 TGFR 베타, 동일 수용체, 그리고 이들의 기능적 단편을 포함한다.
인핸서는 본원에 기술된 조작된 세포에 의해 발현될 수 있고, 조작된 세포의 표면 상에 디스플레이될 수 있거나, 또는 인핸서는 조작된 세포에 의해 주변 세포외 공간으로 분비될 수 있다. 표면 디스플레이 및 분비의 방법들은 당 업계에 잘 알려져있다. 예를 들어, 인핸서는 표면 디스플레이 또는 세포외 공간으로의 분비를 제공하는 펩타이드와의 융합 단백질일 수 있다.
인핸서의 효과는 인핸서 수용체 및 이의 기능적 단편과 같은 추가적인 인자에 의해 보완될 수 있다. 추가적인 인자는 융합 단백질로서 인핸서와 함께 공동 발현되거나 또는 별도의 펩타이드로 발현되어 세포외 공간으로 분비될 수 있다.
하나의 실시 양태에서, 인핸서는 IL-15이다. 이 경우, 추가적인 인자는 IL-15 수용체 및 이의 기능적 단편이다. 기능적 단편은 IL-15 수용체, IL-15RA 및 IL-15RA의 스시 도메인을 포함한다. 적합한 스시 도메인의 하나의 예는 SEQ ID NO. 35를 포함한다. 본 개시내용에 따라, 본원에 개시된 임의의 키메라 항원 수용체 폴리펩타이드는 인간 인터루킨 2 신호 펩타이드 SEQ ID NO. 36을 갖는 인간 인터루킨 15를 포함한다.
인터루킨-15와 그것의 특이적인 수용체 사슬인 IL-15Rα (IL-15-RA)는 NK와 CD8 T 세포를 포함하여 다양한 효과기 세포에서 중요한 기능적 역할을 한다. CD8 + T 세포는 생체내 전이 후에 생존을 유지하기 위해, IL-2, IL-7, IL21 또는 IL-15를 포함하나 이에 한정되지는 않는 자가분비 성장 인자를 발현하도록 변형될 수 있다. 이론에 얽매이는 것을 원하지는 않지만, IL-15는 CD4 결핍을 극복하여 1차 CD8T 세포를 유도하고 기억 CD8 T 세포를 소환할 수 있다고 믿어진다. CD8 T 세포 상에서의 IL-15-RA 또는 IL-15 IL-RA 융합의 과발현은 생체내 및 시험관내에서 그의 생존 및 증식을 유의적으로 증진시킨다. 일부 실시 양태에서, CD4CAR 또는 임의의 CAR은 CAR T 또는 NK의 생존 또는 증식을 증진시키고, 기억 CAR CD8 + T 세포의 팽창을 개선시키기 위해, 모이어티, IL-15, IL15RA 및 IL-15/IL-15R 또는 IL15-RA/IL-15, 또는 이들의 일부 또는 조합 중의 임의의 하나 이상을 발현하는 것을 포함할 수 있다.
환자에서의 암 치료를 위한 CAR T 또는 NK의 생존 또는 지속성 또는 증식을 증진시키기 위해, 본 개시내용은 본원에 기재된 바와 같은 CAR 및 IL-15, IL15RA 및 IL-15/IL-15R 또는 IL15-RA/IL-15, 또는 이의 일부 또는 조합의 모이어티 중 임의의 하나 이상을 갖는 조작된 세포에 관한 것이다.
하나의 실시 양태에서, 조작된 세포는 CD4 키메라 항원 수용체 폴리펩타이드 및 IL-15RA (SEQ ID NO. 1), 및 상응하는 폴리뉴클레오타이드 (SEQ ID NO. 2)를 포함한다.
조작된 세포를 생성하는 방법들
본원에 개시된 임의의 폴리뉴클레오타이드는 조작된 세포 내로 당 업계에 공지된 임의의 방법에 의해 도입될 수 있다.
하나의 실시 양태에서, CAR 폴리뉴클레오타이드는 조작된 세포로 본원에 개시된 임의의 바이러스 벡터에 의해 전달된다.
하나의 실시 양태에서, 증진된 안전성 프로파일 또는 치료 지수를 달성하기 위해, 본원에 개시된 임의의 조작된 세포는 일시적 RNA-변형된 "생물분해성" 버전 또는 이의 유도체 또는 이들의 조합물로서 구축될 수 있다. 본 발명의 RNA-변형된 CAR은 T 세포 또는 NK 세포 내로 전기 천공될 수 있다. 화합물 CAR의 발현은 며칠 동안 지나면서 점차적으로 감소될 수 있다.
본 발명의 일부 실시 양태에서, 본원에 개시된 임의의 조작된 세포는 바이러스 벡터없이 CAR DNA를 숙주 유전체로 통합시키는 트랜스폰슨 시스템 ("슬리핑 뷰티"라고도 함) 내에서 구축될 수 있다.
다중 CAR 단위를 갖는 조작된 세포를 생성하는 방법들
또 다른 실시 양태에서, 본 개시내용은 적어도 2 개의 CAR 단위를 갖는 조작된 세포를 만드는 방법을 제공한다.
일부 실시 양태에서, 다중 CAR 단위는 바이시스트론 (bicistronic) 또는 멀티시스트론 (multicistronic) 발현 벡터를 사용하여 T 또는 NK 세포 내에서 발현된다. 바이시스트론 또는 멀티시스트론 벡터를 구축하기 위해 사용될 수 있는 몇 가지 전략은 비제한적으로, (1) CAR의 오픈 리딩 프레임(open reading frame)에 융합된 다중 프로모터; (2) CAR 단위들 사이의 스플라이싱 신호의 삽입; 그의 발현이 단일 프로모터에 의해 유도되는 CAR의 융합;(3) CAR의 단위들 사이에 단백질분해 절단 부위의 삽입 (자기 절단 펩타이드); 및 (iv) 내부 리보솜 진입 부위 (IRES)의 삽입을 포함한다.
바람직한 실시 양태에서, 다중 CAR 단위는 단일 오픈 리딩 프레임 (ORF) 내에서 발현되어, 이로 인해 다중 CAR 단위를 갖는 하나의 단일 폴리펩타이드를 생성한다. 이 실시 양태에서, 고효율 절단 부위를 함유하는 아미노산 서열 또는 링커가 각각의 CAR 단위 사이에 배치된다.
본원에 사용된 바와 같이, 높은 절단 효율은 번역된 단백질의 50% 초과, 70% 초과, 80% 초과, 또는 90% 초과가 절단되는 것으로 정의된다. 절단 효율은 Kim 2011에 의해 기술된 바와 같이, 웨스턴 블롯 분석으로 측정될 수 있다.
또한, 바람직한 실시 양태에서, 웨스턴 블롯 분석에 도시된 바와 같이 동일한 양의 절단 산물이 존재한다.
고효율 절단 부위의 예는 돼지 테스코바이러스-1 2A (P2A), FMDV 2A (여기에서 F2A로 약칭됨); 에퀸 리니티스A 바이러스(equine rhinitis A virus) (ERAV) 2A (E2A); 및 토세아아시그나 바이러스 (Thoseaasigna virus) 2A (T2A), 세포질 다각체병 바이러스(cytoplasmic polyhedrosis virus) 2A (BmCPV2A) 및 플라세리바이러스(flacherie Virus) 2A (BmIFV2A), 또는 이들의 조합을 포함한다. 바람직한 실시 양태에서, 고효율 절단 부위는 P2A이다. 고효율 절단 부위는 Kim JH, Lee S-R, Li L-H, Park H-J, Park J-H, Lee KY, et al. (2011) High Cleavage Efficiency of a 2A Peptide Derived from Porcine Teschovirus-1 in Human Cell Lines, Zebrafish and Mice. PLoS ONE 6(4): e18556에 기술되어 있고, 그 내용은 본원에 참조로 포함된다.
다중 CAR 단위가 단일 오픈 리딩 프레임 (ORF)에서 발현되는 실시 양태들에서, 발현은 강력한 프로모터의 제어하에 있다. 강력한 프로모터들의 예는 SFFV 프로모터 및 이의 유도체를 포함한다.
CAR
폴리펩타이드
및
인핸서를
갖는 조작된 세포
또 다른 실시 양태에서, 본 개시내용은 적어도 하나의 CAR 단위 및 인핸서를 발현하는 조작된 세포를 제조하는 방법을 제공한다.
일부 실시 양태에서, 적어도 하나의 CAR 단위 및 인핸서는 바이시스트론 (bicistronic) 또는 멀티시스트론 (multicistronic) 발현 벡터를 사용하여 T 또는 NK 세포 내에서 발현된다. 바이시스트론 또는 멀티시스트론 벡터를 구축하기 위해 사용될 수 있는 몇 가지 전략은, 이에 제한되는 것은 아니지만, (1) CAR의 오픈 리딩 프레임에 융합된 다중 프로모터; (2) CAR 단위들 사이에 스플라이싱 신호 삽입; 그의 발현이 단일 프로모터에 의해 유도되는 CAR의 융합; (3) CAR의 단위들 사이에 단백질분해의 절단 부위의 삽입 (자기 절단 펩타이드); 및 (4) 내부 리보솜 진입 부위 (IRES)의 삽입을 포함한다.
바람직한 실시 양태에서, 적어도 하나의 CAR 단위 및 인핸서가 단일 오픈 리딩 프레임 (ORF)에서 발현되어, 이에 의해 적어도 하나의 CAR 단위 및 인핸서를 갖는 단일 폴리펩타이드를 생성한다. 이 실시 양태에서, 고효율 절단 부위를 함유하는 아미노산 서열 또는 링커는 각 CAR 단위 사이 및 CAR 단위와 인핸서 사이에 배치된다. 이 실시 양태에서, ORF는 강력한 프로모터의 제어하에 있다. 강력한 프로모터의 예는 SFFV 프로모터 및 이의 유도체를 포함한다.
또한, 바람직한 실시 양태에서, 웨스턴 블롯 분석에 도시된 바와 같이, 동일한 양의 절단 산물이 존재한다.
본원에 개시된 조성물을 사용하는 치료 방법
또 다른 실시 양태에서, 본 발명은 암 치료에서 동종이계 이식 이전에 조건화 (conditioning)를 위해 CD45를 표적화하는 방법을 제공한다. CD45는 백혈구 공통 항원 (LCA)으로도 알려져 있으며 적혈구와 혈소판을 제외한 사실상 모든 조혈 기원 세포에서 발현되는 티로신 포스파타제이다. 대부분의 혈액 악성 종양은 CD45를 발현한다. 예를 들어, 85% 내지 90%의 급성 림프성 및 골수성 백혈병은 CD45를 발현한다. CD45는 비조혈 기원에서는 발견되지 않는다. 또한, CD45는 악성 세포 및 백혈구에서 세포 당 약 200,000 분자의 고밀도 평균 카피수로 발현된다. CD45는 다양한 혈액 악성 종양에 대해 이상적인 표적을 제시한다. 그러나, CAR T 및 NK 세포는 또한 CD45를 발현한다. 내인성 CD45의 불활성화가 없다면, CD45를 표적으로 하는 CAR로 무장한 CAR T 또는 NK 세포는 자가-살상을 일으킬 수 있다.
TCR 복합체와의 CD45의 회합은 항원에 반응하여 T 세포 활성화를 조절하는 것에 필수적이다. CD45 결핍 T 세포가 항원을 제시할 수 없다는 것은 T 세포 수용체 (TCR)를 통한 감소된 신호전달 때문이다. TCR은 항원 제시에 반응하는 T 세포의 활성화에 필수적인 역할을 하는 세포 표면 수용체이다. TCR은 일반적으로 두 개의 사슬, 알파 및 베타로부터 제조되는 데 그들은 형질도입 하위단위인 CD3와 회합되어 세포 표면에 존재하는 T 세포 수용체 복합체를 형성한다.
본 발명의 다중 CAR (화합물 CAR, cCAR)은 암세포가 CAR 활성에 저항하는 주요 메커니즘 즉, 암세포 표면으로부터의 표적 항원의 하향조절 또는 이종 발현을 퇴치한다는 것이 놀랍게도, 발견되었다. 이 메커니즘은 암세포가 CAR 치료법으로부터 "숨겨 지도록"하는 데 이 현상을 '항원 회피'라고 한다. 본 개시내용은 종양을 신속하게 제거하기 위해 2개 이상의 항원의 조합을 인식함으로써 암 항원 회피를 미연에 방지한다.
본 발명은 cCAR을 이용한 다수 항원의 동시 표적화 방법을 제공하여 표적 항원 손실 또는 하향 조절에 기초한 종양 선택의 가능성을 최소화함으로써 개선된 종양 제어의 결과를 낳는다.
개시된 본 발명은 종양 세포에 존재하는 상이한 또는 동일한 표면 항원을 표적으로 하는 T 또는 NK 세포에서의 화합물 (다중 또는 화합물) cCAR을 포함한다. 본 발명의 화합물 키메라 항원 수용체는 링커에 의해 연결된 적어도 다수의 키메라 수용체 구축물을 포함하고 동일하거나 상이한 항원을 표적으로 한다. 예를 들어, 화합물 CAR (cCAR) 구축물에 존재하는 CAR 구축물 각각은 항원 인식 도메인, 세포외 도메인, 막통과 도메인 및/또는 세포질 도메인을 포함한다. 세포외 도메인 및 막통과 도메인은 이러한 도메인에 대한 임의의 목적하는 공급원으로부터 유래될 수 있다. 다중 CAR 구축물은 링커에 의해 연결된다. 화합물 CAR 구축물의 발현은 프로모터에 의해 유도된다. 링커는 단백질 또는 펩타이드가 생성된 후에 자가 분해되는 펩타이드 또는 단백질의 일부일 수 있다 (자체-절단되는 펩타이드라고도 불림).
하나의 실시 양태에서, 본 발명의 화합물 CAR은 골수이형성증후군 및 급성 골수성 백혈병 (AML) 집단을 표적으로 한다. 골수이형성증후군 (MDS)은 고령자 중에서 가장 흔히 발생하는 난치성 조혈 줄기 세포 악성 종양이고 미국에서 매년 약 14,000 건의 새로운 케이스를 갖는다. MDS 케이스의 약 30-40 %가 AML로 진행된다. MDS의 발생은 우리의 인구 고령화에 따라 계속하여 증가하고 있다. 비록 MDS와 AML이 열심히 연구되고 있지만, 만족할만한 치료법은 개발되어 있지 않다.
본 발명의 조성물 및 방법은 암치료, 특히 폐암, 흑색종, 유방암, 전립선암, 대장 암, 신장세포암종, 난소암, 뇌암, 육종, 백혈병 및 림프종 치료에서의 면역 요법에 사용하기 위한 1차 및 보조 자극 신호 모두를 전달하는 T 림프구 또는 NK 세포 집단을 생성하는 데 사용될 수 있다.
면역 요법은 일반적으로 면역 효과기 세포 및 분자의 사용에 의존하여 암세포를 표적화하고 파괴한다. 효과기는 직접 또는 간접적으로 종양 세포 표적과 상호 작용하는 표면 분자를 가지는 림프구일 수 있다. 다양한 효과기 세포는 세포독성 T 세포, NK 세포 및 NK-92 세포를 포함한다. 본 발명에서 기술된 조성물 및 방법은 화학 요법, 수술, 방사선, 유전자 요법 등과 같은 다른 유형의 암 요법과 함께 이용될 수 있다. 본 발명에서 기술된 조성물 및 방법은 염증, 면역 질환 및 감염성 질환과 같은 면역 반응에 의존하는 다른 질환 상태에 이용될 수 있다.
일부 실시 양태에서, 본 발명의 화합물 CAR은 최소 잔류 질환을 갖고 더 이상 화학 요법에 더 이상 반응하지 않는 환자에 대한 완전한 완화를 달성함으로써 골수 이식에 대한 가교 역할을 할 수 있다. 또 다른 실시 양태에서, 화합물 CAR은 백혈병 세포를 제거하고, 이어서 백혈구 감소증을 지원하기 위해 골수 줄기 세포를 구출한다.
일부 실시 양태에서, 본 개시내용의 화합물 CAR은 표적 항원의 하향 조절에 의해 암세포가 CAR 활성에 저항하는 주요 메커니즘을 퇴치할 수 있다. 또 다른 실시 양태에서, 본 발명의 화합물 CAR은 또한 표준 CAR T/NK 세포 요법에서 상당한 어려움을 발생시키는 암세포의 이질성을 퇴치할 수 있다. 추가의 실시 양태에서, 개시된 화합물 CAR은 제1 CAR이 벌크 종양 집단을 표적으로 하고 또 다른 CAR이 암 재발을 피하기 위해 암 또는 백혈병 줄기 세포를 근절시키도록 설계된다.
하나의 실시 양태에서, 본 개시내용은 CD33 항원 또는 CD123 항원, 또는 둘 다를 갖는, CD33 항원 인식 도메인을 갖는 키메라 항원 수용체 폴리펩타이드 및 CD123 항원 인식 도메인을 갖는 키메라 항원 수용체 폴리펩타이드 중에서 적어도 하나를 가지는 조작된 세포와 접촉시킴으로써 상기 세포를 제거하는 방법을 제공한다.
CD33 항원 및 CD123 항원 중 적어도 하나를 갖는 세포는 급성 골수성 백혈병, 전구체 급성 림프모구성 백혈병, 만성 골수증식성 신생물, 만성 골수성 백혈병, 골수이형성증후군, 모세포성 형질세포양 수지상 신생물 (BPDCN), 호지킨 림프종, 비만 세포증 및 모발상세포 백혈병 세포를 포함한다.
또 다른 실시 양태에서, 본 개시내용은 조혈 줄기 세포 이식을 위한 골수절제 조건화 요법 (myeloablative conditioning regimens)을 제공하는 방법을 제공한다. 이 실시 양태에서, CD33 단위 및 CD123 단위를 갖는 조작된 T 또는 NK 세포는 이를 필요로 하는 환자에게 투여된다.
추가의 실시 양태에서, 본 개시내용은 CD123 또는 CD33, 또는 이들 모두를 발현하는 백혈병 줄기 세포 (LSC) 또는 벌크 백혈병 세포를 근절 또는 살상시키는 방법을 제공한다. 이 실시 양태에서, CD33 단위 및 CD123 단위를 갖는 조작된T 또는 NK 세포는 이를 필요로 하는 환자에게 투여된다.
추가의 실시 양태에서, T 또는 NK 세포에서의 화합물 CAR은 CD34+ CD38- 백혈병 줄기 세포 또는 CD123 또는 CD33 또는 이들 모두를 발현하는 벌크 백혈병 세포를 근절시키거나 살상시키는데 사용될 수 있다.
일부 실시 양태에서, 화합물 CAR은 CD19 또는 CD20 항원 또는 둘 다를 발현하는 세포를 표적으로 한다. 또 다른 실시 양태에서, 화합물 CAR은 CD19 또는 CD22 항원 또는 둘 다를 발현하는 세포를 표적으로 한다. 표적화 세포는 비제한적으로 B 세포 림프종 또는 백혈병과 같은 암 세포일 수 있다. 추가의 실시 양태에서, 표적 항원은 ROR1, PSMA, MAGE A3, 당지질, 글리피칸 3, F77, GD-2, WT1, CEA, HER-2/neu, MAGE-3, MAGE-4, MAGE-5, MAGE- 6, 알파태아단백, CA 19-9, CA 72-4, NY-ESO, FAP, ErbB, c-Met, MART-1, CD30, EGFRvIII, 면역글로불린 카파 및 람다, CD38, CD52, CD3, CD4, CD8, CD5, CD7, CD2, 및 CD138의 그룹 중 적어도 하나를 포함할 수 있지만 이에 국한되지는 않는다. 표적 항원은 또한 인간 유두종 바이러스 (HPV) 또는 EBV (엡스타인 바 바이러스) 항원으로부터의 E6 및 E7과 같은 바이러스 또는 진균 항원을 포함할 수 있다.
일부 실시 양태에서, 화합물 CAR은 CD19 또는 CD123 항원 또는 둘 다를 발현하는 세포를 표적으로 한다. 표적화 세포는 비제한적으로 B 세포 림프종 또는 백혈병과 같은 암세포이다.
추가의 실시 양태에서, 화합물 CAR은 CS1 및/또는 B 세포 성숙 항원 (BCMA) 또는 둘 다를 발현하는 세포를 표적으로 한다. 또 다른 실시 양태에서, 표적화 세포는 비제한적으로 다발성 골수종과 같은 악성 형질 세포이다.
일부 실시 양태에서, 화합물 CAR은 CS1, BCMA, CD267, BAFF-R, CD38, CD138, CD52, CD19, CD20, 인터루킨 6 수용체 및 NY-ESO-1 항원을 포함하나 이에 한정되지는 않는 다수의 항원을 발현하는 세포를 표적으로 한다. 또 다른 실시 양태에서, 표적화 세포는 비제한적으로 다발성 골수종과 같은 악성 형질 세포이다.
일부 실시 양태에서, 화합물 CAR은 알파 태아단백 (AFP) 및 글리피칸-3 (GPC3)을 포함하나 이에 한정되지 않는 다수의 항원을 발현하는 세포를 표적으로 한다. 또 다른 실시 양태에서, 표적화 세포는 간세포 암종, 섬유층판암종, 간모세포종, 간의 미분화 배아 육종 및 간엽성 과오증, 폐-편평 세포 암종, 고환 비정상피종성 생식세포종양, 지방육종, 난소 및 생식샘외 난황낭종, 난소 융모막암종, 기형종, 난소 투명세포 암종 및 태반 부위 융모성 종양이다.
본 발명에 따르면, 상이한 또는 동일한 항원을 표적으로 하는 화합물 CAR을 포함하는 T 또는 NK 세포는 종양 회피를 상쇄시키고 종양 세포의 동시 표적화를 가능하게 한다.
본원에 개시된 화합물 CAR을 포함하는 T 또는 NK 숙주 세포는 본 개시내용에서 실시된다. 화합물 CAR의 뉴클레오타이드 및 폴리펩타이드 구축물, 서열, 숙주 세포, 벡터는 본 개시내용의 일부로 간주되고 본원에서 실시된다.
일부 실시 양태에서, 화합물 CAR은 현재 개발되고 있거나 또는 시장에서 이용가능한 임의의 화학 요법제와 조합하여 투여된다. 일부 실시 양태에서, 화합물 CAR은 혈액 악성종양, 암, 비혈액 악성종양, 염증성 질환, HIV 및 HTLV와 같은 감염성 질환 및 기타를 포함하나 이에 한정되지는 않는 질환에 대한 1차 치료로서 투여된다. 하나의 실시 양태에서, 화합물 CAR을 발현하는 T 세포는 적응 면역요법으로서 동일하거나 상이한 화합물 CAR을 발현하는 NK 세포와 함께 공동투여된다. 화합물 CAR NK 세포는 세포를 표적화하는 신속한 선천성 활성을 제공하는 반면 화합물 T 세포는 상대적으로 오래 지속되는 적응 면역 활성을 제공한다.
하나의 실시 양태에서, 화합물 CAR을 발현하는 세포는 포유동물, 예를 들어, 화학 요법에 내성이 있고 골수 줄기 세포 이식에 적합하지 않은 환자를 위한 골수 줄기 이식의 가교로서 투여된다.
일부 실시 양태에서, 화합물 CAR은 트랜스진을 공동발현시키고, 표적화 종양 병변에서의 IL-12와 같은 트랜스진 생성물을 방출하고 종양 미세환경을 추가로 조절한다.
한 실시 양태에서, 화합물 CAR을 발현하는 세포는 질환에 대한 치료의 일부로서 골수 골수성 제거를 위해 포유동물에 투여된다.
특정 실시 양태에서, 화합물 CAR을 발현하는 세포는 포유동물, 예를 들어, 인간에게 투여되는 T 세포 또는 NK 세포일 수 있다. 본 개시내용은 화합물 CAR의 투여에 의해 장애 또는 질환을 갖는 포유동물을 치료하는 방법을 포함한다. 표적화 세포는 암세포, 또는 감염성 질환, 염증 및 자가면역 장애와 같은 임의의 다른 질환 상태에 의해 영향을 받은 세포일 수 있다.
본 발명은 화합물 CAR의 단편, 돌연변이 또는 변이체 (예를 들어, 변형된 형태) 또는 T/NK 세포의 자극 및 증식을 유도하는 능력을 보유하는 항원의 용도를 포함하는 것으로 의도된다. "단백질의 형태"는 적어도 하나의 CAR 또는 항원과 유의한 상동성을 공유하고 T/NK 세포의 자극 및 증식을 일으킬 수 있는 단백질을 의미하는 것으로 의도된다. 본원에서 사용되는 용어 "생물학적 활성"또는 "단백질의 생물학적 활성 형태"는 세포의 항종양 활성을 일으킬 수 있는 단백질 또는 변종의 형태를 포함하는 것을 의미한다.
본 발명의 조성물 및 방법은 암치료, 특히 폐암, 흑색종, 유방암, 전립선암, 대장암, 신장세포암종, 난소암, 신경모세포종, 횡문근육종, 백혈병 및 림프종 치료에서의 면역 요법에 사용하기 위한 1차 및 보조 자극 신호 둘 다를 전달하는 T/NK 세포 집단을 생성하는 데 사용될 수 있다. 본 발명에서 기술된 조성물 및 방법은 화학 요법, 수술, 방사선, 유전자 요법 등과 같은 다른 유형의 암 요법과 함께 이용될 수 있다.
일부 실시 양태에서, 본 발명은 CAR 또는 화합물 CAR T 세포 또는 NK 세포를 자가면역 질병을 가진 환자에게 투여하는 것에 의해 자가면역 질환을 가진 환자에서의 B 세포, 미성숙 B 세포, 기억 B 세포, 형질모세포, 오래 생존한 형질세포, 또는 형질세포를 고갈시키는 방법을 개시한다. CAR 표적화 세포는 항원 BCMA, TACI 및 BAFF-R 중 하나 또는 둘 또는 전부를 발현하는 B 또는 형질 세포이다. 자가면역 질환은 전신피부경화증, 다발경화증, 건선, 피부염, 염증성 장질환 (예컨대, 크론병 및 궤양성 결장염), 전신 홍반성 루푸스, 혈관염, 류마티스관절염, 쇼그렌증후군, 다발성근육염, 육아종증 및 혈관염, 애디슨병, 항원-항체 복합체 매개 질환 그리고 항사구체 기저막 질환을 포함한다.
다수 세포외 세포 마커는 종양-연관 항원으로서의 가치, 및 이에 따라 CAR T/NK 세포 요법을 위한 잠재적 표적으로서 지금 연구되고 있다. 그러나, 온-표적 (on-target), 오프-종양 (off-tumor) 유해사건을 야기하는 건강한 조직상에서의 이들 항원의 발현은 오프-표적 독성 이외에 주요 안전 문제로 남아있다. 또한, CAR T/NK 세포 요법의 주요 한계는 종양발생에 필수적이지 않은 분자를 표적으로 할 때 항원 회피 변이체에 대해 선택할 가능성에 있다. 따라서, 표적 항원의 발현이 거의 또는 전혀 없는 상태를 지속하는 악성 세포는 CAR T/NK 세포의 높은 친화성 작용에도 불구하고 그들의 세포를 회피할 수 있다.
본 발명에 따르면, 자연 살상 세포 (NK 세포)는 CAR 유도된 살상에 대한 대체 세포독성 효과기를 나타낸다. T 세포와 달리, NK 세포는 사전 활성화를 필요로 하지 않으며 본질적으로 세포용해 기능을 나타낸다. NK 세포에서의 cCAR의 추가 발현은 NK 세포가 암, 특히 NK 세포 치료에 저항성인 암세포를 효과적으로 살상할 수 있게 한다.
또한, NK 세포는 이식편-대-숙주 질환 (GvHD)을 유도하는 위험없이 항암 효과를 매개하는 것으로 알려져있다.
연구에 의하면 CD34+ CD38-AML 세포상에서 CD123의 비정상적인 과발현이 나타난 반면, 정상 골수 대응부 CD34+ CD38-에서는 CD123이 발현되지 않았다 (Jordan, Upchurch 등, 2000). 이들 CD123 +, CD34+ CD38- 집단은 이들 세포가 백혈병 과정을 면역결핍 마우스에서 개시하고 이를 유지할 수 있기 때문에 LSC로 간주되어 왔다.
CD34+ /CD38- /CD123 + LSC의 수는 AML 환자의 임상 결과를 예측하는 데 사용될 수 있다. AML 환자 중에서 15% 초과의 CD34+ / CD38- /CD123+ 세포는 완전 완화의 결여 및 바람직하지 않은 세포유전학적 프로파일과 관련이 있다. 또한, 1% 초과의 CD34+ /CD38- /CD123+ 세포의 존재는 무질환 생존 및 전반적인 생존에 부정적인 영향을 줄 수 있었다.
현재, MDS와 AML을 위한 요법은 백혈병 모세포에 초점을 맞추고 있는데 이는 이들이 매우 풍부하고 환자에 대한 가장 직접적인 문제를 명확하게 나타내기 때문이다. 중요하게도, 백혈병 줄기 세포 (LSC)는 대부분의 다른 백혈병 세포 ("모"세포)와 다소 다르며, 그들은 드문 하위 집단을 구성한다. 모세포를 살상하는 것은 단기적인 완화를 제공할 수 있지만, LSC가 만약 파괴되지 않으면 항상 다시 자라나서 환자의 재발을 초래할 것이다. MDS 질환에 대한 내구성 있는 치료를 달성하기 위해서는 LSC가 반드시 파괴되어야 한다. 불행하게도, 표준 약물 요법은 MDS 또는 AML LSC에 효과적이지 않다. 따라서 백혈병 줄기 세포 집단과 벌크 백혈병 집단 모두를 특이적으로 표적으로 할 수 있는 새로운 요법을 개발하는 것이 중요하다. 본 발명에서 개시된 화합물 CAR은 이들 집단 모두를 표적으로 하며, 본원에서 실시된다.
본 발명에 따르면, 놀랍게도, 치료를 위한 동종이계 생성물로서 사용될 수 있는 기성품을 NK 세포가 제공한다는 것이 밝혀졌다. 따라서, 본 발명에 따르면, cCAR 세포 요법은 현재의 최신 기술이 요구하는 바와 같이 환자 특이적 기반상에서 수행되는 것이 필요하다. 본 발명의 출원인은 효과적인 CAR 세포 기반 요법을 위해 환자의 림프구 또는 종양 침윤된 림프구가 단리될 필요가 없는 새로운 면역 요법을 발견하였다.
동종이계 또는 자가 NK 세포는 빠른 면역 반응을 유도하는 것으로 예상되지만 그들의 제한된 수명으로 인해 혈액순환으로부터 상대적으로 빠르게 사라진다. 따라서, 본 출원인은 cCAR 세포 기반 요법을 사용하여 지속되는 부작용에 대한 우려가 감소됨을 놀랍게도 발견했다.
본 발명의 한 양태에 따르면, NK 세포는 팽창되고 본 발명에 따른 cCAR로 형질 감염될 수 있다. NK 세포는 제대혈, 말초 혈액, iPS 세포 및 배아 줄기 세포로부터 유래될 수 있다. 본 발명의 한 양태에 따르면, NK-92 세포는 팽창되고 cCAR로 형질 감염될 수 있다. NK-92는 자연 살상 세포 (NK 세포)의 특색과 특징을 지닌 지속적으로 성장하는 세포주이다. NK-92 세포주는 IL-2 의존적이며 안전하고 실현 가능한 것으로 입증되었다. cCAR 발현 NK-92 세포는 피더세포 (feeder cell)와의 공동 배양 유무와 관계없이 무혈청- 배지에서 팽창될 수 있다. 관심 cCAR을 운반하는 NK-92의 순수한 집단은 분류에 의해 얻을 수 있다.
적절한 표면 표적 항원의 식별은 적응 면역 요법에서 CAR T/NK 세포를 발생시키기 위한 전제 조건이다.
본 발명의 한 양태에서, CD123 항원은 cCAR 요법의 표적 중 하나이다. 인터루킨 3 수용체의 알파 사슬인 CD123은 급성 골수성 백혈병 (AML), B 세포 급성 림프모구성 백혈병 (B-ALL), 모발상세포 백혈병 및 모세포성 형질세포양 수지상 신생물을 비롯한 다양한 혈액 악성 종양에서 과발현된다. CD123은 정상 조혈 줄기 세포에서 부재하거나 최소한으로 발현된다. 더 중요하게, CD123은 백혈병 줄기 세포 (LSC)와 관련된 백혈병 세포의 하위세트 상에서 발현되며, 이것의 제거는 질환의 불응성 및 재발을 방지하는 데 필수적이다.
본 발명의 한 양태에서, CD33 항원은 cCAR 요법을 위한 표적 중 하나이다. CD33은 급성 골수성 백혈병에서 90%의 악성 세포에서 발현되는 막통과 수용체이다. 따라서, 본 발명에 의하면, CD123 및 CD33 표적 항원은 특히 안전의 관점에서 매력적이다.
본 발명에 따라서, 화합물 CD33CD123 CAR은 만성 골수성 백혈병 (CML) 집단의 치유적 치료에 매우 효과적일 수 있다. 만성 골수성 백혈병 (CML)에는 CD34+CD38-인 세포의 드문 하위세트가 있다. 이 집단은 LSC로 구성된다고 간주된다. 증가된 수의 LSC는 질환의 진행과 관련이 있다. 소분자 Bcr-Abl 티로신 키나아제 억제제 (TKI)는 CP-CML 환자의 전반적인 생존을 상당히 개선시키는 것으로 나타난다. 그렇지만, LSC는 TKI 요법에 저항성이 있는 것으로 생각된다. CML 저항성 LSC를 표적으로 하는 새로운 요법은 CML 치료에 시급히 필요하며 본 발명에 개시된 화합물 CD33CD123 CAR에서 새로운 요법이 실시된다. CD123은 CD34+CD38- 집단에서 높게 발현된다. 본 발명에 따라서, 화합물 CD33CD123 CAR은 이 집단의 치유적 치료에 대해 매우 효과적이다.
본 발명의 하나의 실시 양태에서, cCAR에서의 CD123 및 CD33 둘 모두를 발현하는 백혈병 세포가 치유적 치료로서 사용된다. CD33은 골수계, 골수성 백혈병 모세포 및 성숙 단핵구의 세포 상에서 발현되지만 정상 다능성 조혈 줄기 세포에서는 발현되지 않는다 (Griffin, Linch 등, 1984). CD33은 CML, 골수 증식성 신생물 및 MDS에서의 백혈병 세포에서 광범위하게 발현된다.
급성 골수성 백혈병 (AML) 환자의 상당수가 표준 화학치료 요법에 불응성이거나, 치료 후 질환 재발을 경험함에 따라 (Burnett 2012), AML에 대한 CAR T 세포 면역 요법의 개발은 상당한 임상적 필요성을 해결할 잠재력을 가진다. 이들 환자의 대다수에서, 백혈병 세포는 CD123 및 CD33 둘 모두를 발현하며, 본원에 개시된 화합물 CD33CD123 CAR에 광범위한 임상적 적용성을 부여한다. 따라서, 본 발명은 다수 백혈병-연관 항원을 표적으로 하는 다중 CAR을 포함하는 새로운 다중 cCAR T/NK 세포 구축물을 개시하여, 항원 회피 메커니즘을 상쇄시키고 보조 자극 도메인 활성화의 상승작용적 효과에 의해 백혈병 줄기 세포를 비롯한 백혈병 세포를 표적으로 하여, 보다 강력하고 안전하며 효과적인 요법을 제공한다.
본 발명은 표적 항원을 공동 발현하는 세포에 대한 항-종양 활성의 증진된 효력을 지니면서도 한개 항원만을 발현하는 종양 세포에 대한 감수성을 보유하는 화합물 CAR 구축물을 추가로 개시한다. 또한, 화합물 CAR의 각 CAR은 1개 또는 2개의 보조 자극 도메인 및 특정 표적의 존재 하에서의 강력한 살상 능력을 포함한다.
유방암과 상피성 난소암을 포함하는 고형 종양을 표적으로 하는, 트랜스-시그널링 (trans-signaling) CAR의 이중특이성(dual specificity)에 대한 전임상 연구에서 CD3ζ 세포내 신호전달 도메인은 제2세대 CAR 유래의 보조 자극 도메인으로부터 분리되어 있다. 바꿔 말하면, 하나의 CAR은 보조 자극 도메인이 없는 제1세대 CAR을 포함하고 또다른 CAR는 CD3제타 세포내 도메인이 없다. 따라서 T 세포 활성화와 강력한 살상을 위해서는 표적 항원 둘 다의 존재가 요구된다. 따라서, 이들은 감수성을 감소시키는 대신에, 표적 특이성을 증가시키면서, 2개의 표적 항원 중 1개의 건강한 조직 발현에 의해 유발되는 오프-종양 독성을 감소시키는 방법으로서 제안되었다. 하나의 실시 양태에서, 화합물 CAR은 화합물 CD123CD19 CAR이다. 집단의 하위세트에서 90% 초과의 B-ALL이 CD123을 발현한다는 것이 밝혀졌다. AML 및 MDS와 마찬가지로, 희귀한 LSC 집단이 B-ALL에 존재하는 것으로 간주되었다. 따라서, 본 발명에 따라 백혈병 줄기 세포 및 벌크 백혈병 집단 둘 다를 표적으로 하는 것은 B-ALL에 적용될 수 있다. 본 발명대로, CD19가 B-세포 림프구 집단의 상이한 단계에서 광범위하게 발현됨에 따라 B-ALL에서 발현되는 CD123 및 CD19 표면 항원은 표적이 될 수 있다.
다발성 골수종 (MM)은 미국에서 두 번째로 제일 흔한 혈액 악성 종양이며 골수 또는 골수외 부위에 축적된 클론 형질세포로부터 유래한다. MM은 약 4.5 년의 중앙 생존 기간을 갖는 난치병이다 (Kumar, Rajkumar 등. 2008). 전임상 개발에 있어서 항-골수종 CAR이 개발되었고 CAR 표적은 CD38, CS1, B 세포 성숙 항원 (BCMA) 및 CD38을 포함한다. 그러나, 표면 항원 발현의 이질성은 통상적으로 악성 형질 세포에서 발생하며 (Ruiz-Arguelles 및 San Miguel 1994), 이는 CAR에 대한 어려운 표적이 되게 한다. 악성 형질 세포는 또한 낮은 수준의 CD19를 발현한다. 이전에, 골수종 줄기 세포도 CD19를 포함하는 일부 B 세포 마커를 발현한다는 것이 밝혀졌다. 이 집단을 표적으로 하는 것은 표준 및 다른 골수종 CAR 요법과 함께 골수종 치료에 효과적일 수 있다.
다발성 골수종 (MM)은 형질 세포의 클론 팽창을 갖는 혈액 악성 종양이다. 치료에서의 중요한 진전에도 불구하고, 골수종은 여전히 난치병으로 남아 있다; 따라서 새로운 치료적 접근이 시급히 필요하다.
CS1 (CD319 또는 SLAMF7이라고도 함)은 SLAMF7 유전자에 의해 코딩되는 단백질이다. 표면 항원 CS1은 정상적인 형질 세포 및 골수종 세포 (악성 형질 세포)에 대한 강한 마커이다.
B 세포 성숙 항원 (BCMA) 또는 CD269라고도 지칭되는 종양 괴사 인자 수용체 수퍼패밀리의 멤버 17 (TNFRSF17)은 형질 세포 및 악성 형질 세포의 말기에서 거의 독점적으로 발현된다. 그의 발현은 다른 조직에는 나타나지 않으며, 이는 CAR T 또는 NK 세포를 위한 표적으로서의 가능성을 나타낸다.
악성 형질 세포는 CD269 및 CS1에 대해 다양한 정도의 항원 이질성을 나타낸다. CD269 또는 CS1을 표적으로 하는 단일 CAR 단위 제품은 벌크 종양에서의 대다수 세포를 표적으로 할 수 있어서 초기 강한 항-종양 반응을 초래한다. 이어서, 잔존하는 희귀한 비-표적화 세포는 팽창되고 질환 재발을 일으킨다. 다발성 골수종은 특히 이질적이지만 이 현상은 다른 백혈병이나 종양에도 확실하게 적용될 수 있다. BCMA CAR T 세포를 이용한 NIH에서의 최근 임상 시험은 다발성 골수종을 가진 일부 환자에서 완전한 반응을 보이는 유망한 결과를 보여주었다. 그러나, 이들 환자는 17주 후에 재발했는데 이는 항원 회피 때문일 수 있다. 항원 회피는 CD19 CAR 및 NY-ESO1 CAR T 세포 치료에서도 나타난다. 따라서, 재발을 막기 위해서는 더욱 효과적인 CAR T 세포 치료가 긴급히 필요하다.
본 발명의 한 양태에서, BCMA 및 CS1은 BCMACS1 CAR 요법의 표적이다.
일부 실시 양태에서, 화합물 CAR은 BCMA 또는 CS1 항원 또는 둘 다를 발현하는 세포를 표적으로 한다. 표적화 세포는 비제한적으로 림프종, 또는 백혈병 또는 형질 세포 신생물과 같은 암세포일 수 있다. 추가의 실시 양태에서, 형질 세포 신생물은 형질 세포 백혈병, 다발성 골수종, 형질 세포종, 중쇄 질환, 아밀로이드증, 발덴스트롬 마크로글로불린혈증, 중쇄 질환, 고립성 골 형질세포종, 미확정 단클론 감마글로불린혈증(MGUS) 및 무증상 다발성 골수종으로부터 선택된다.
BAFF (B 세포 활성화 인자) 및 APRIL (증식-유도성 리간드)은 고친화도로 TACI (TNFRSF1 3B 또는 CD267이라고도 함) 및 BCMA와 특이적으로 결합하는 두 개의 TNF 동족체이다. BAFF (BLyS라고도 공지됨)는 BAFF-R에 결합하고 B 세포의 후기 단계의 생존 및 증식을 증진시키는 데 기능적으로 관여한다. BAFF는 일부 자가면역 장애를 동반하는 것으로 나타났다. APRIL은 항체 클래스 전환의 증진에 중요한 역할을 한다. BAFF와 APRIL 둘 다는 악성 형질 세포에 대한 성장 및 생존 인자로서 연루되어왔다.
악성 형질 세포에서의 리간드-수용체 상호 작용은 도 45에서 설명된다.
일부 실시 양태에서, 화합물 CAR은 TACI 또는 CS1 항원 또는 둘 다를 발현하는 세포를 표적으로 한다. 또 다른 실시 양태에서, 화합물 CAR은 TACI 또는 CS1 항원 또는 둘 다를 발현하는 세포를 표적으로 한다. 표적화 세포는 비제한적으로 림프종, 또는 백혈병 또는 형질 세포 신생물과 같은 암세포일 수 있다. 추가의 실시 양태에서, 형질 세포 신생물은 형질 세포 백혈병, 다발성 골수종, 형질 세포종, 중쇄 질환, 아밀로이드증, 발덴스트롬 마크로글로불린혈증, 중쇄 질환, 고립성 골 형질세포종, 미확정 단클론 감마글로불린혈증(MGUS) 및 무증상 다발성 골수종으로부터 선택된다. 표적 세포는 또한 B 세포 중 1개 또는 2개 또는 다수의 상이한 세포 유형, 미성숙 B 세포, 나이브 B 세포, 중심 모세포, 중심 세포, 기억 B 세포, 형질 모세포, 오래 생존한 형질 세포, 형질 세포일 수 있다. 이들 세포는 전신피부경화증, 다발성경화증, 건선, 피부염, 염증성 장질환 (예컨대, 크론 병 및 궤양성 결장염), 전신 홍반성 루푸스, 혈관염, 류마티스 관절염, 쇼그렌증후군, 다발성근육염, 육아종증 및 혈관염, 애디슨 병, 항원 - 항체 복합체 매개 질환, 및 항 - 사구체 기저막 질환을 포함하는 자가 면역 질환과 연관된다.
일부 실시 양태에서, 화합물 CAR은 BAFF-R 또는 CS1 항원 또는 둘 다를 발현하는 세포를 표적으로 한다. 또 다른 실시 양태에서, 화합물 CAR은 BAFF-R 또는 CS1 항원 또는 둘 다를 발현하는 세포를 표적으로 한다. 표적화 세포는 림프종, 또는 백혈병 또는 형질 세포 종양과 같은, 그러나 이에 한정되지 않는 암세포일 수 있다. 추가의 실시 양태에서, 형질 세포 종양은 형질 세포 백혈병, 다발성 골수종, 형질 세포종, 중쇄 질환, 아밀로이드증, 발덴스트롬 마크로글로불린혈증, 중쇄 질환, 고립성 골 형질세포종, 미확정 단클론 감마글로불린혈증(MGUS) 및 무증상 다발성 골수종으로부터 선택된다.
일부 실시 양태에서, 화합물 CAR (cCAR)은 BAFF-R, BCMA, TACI 및 CS1 항원 중 1개 또는 2개 또는 모두를 발현하는 세포를 표적으로 한다.
일부 실시 양태에서, cCAR에서의 CAR의 단위는 1) BAFF-R, BCMA, TACI 및 CS1 중 어느 하나에 대한 scFv; 2) 힌지 영역; 3) 보조 자극 도메인(들) 및 세포내 신호전달 도메인을 포함할 수 있다.
일부 실시 양태에서, cCAR에서의 CAR의 단위는 1) BCMA 또는 TACI 또는 BAFF-R 결합 도메인, 또는 APRIL 결합 도메인; 2) 힌지 영역; 3) 보조 자극 도메인(들) 및 세포내 신호전달 도메인을 포함할 수 있다.
추가의 실시 양태에서, BCMA 또는 TAC1 또는 BAFF-R 결합 도메인은 APRIL 및 BAFF 분자의 일부 또는 전부일 수 있다.
일부 실시 양태에서, cCAR에서의 CAR의 단위는 1) BCMA 또는 CS1에 대한 scFv; 2) 힌지 영역; 3) 보조 자극 도메인(들) 및 세포내 신호전달 도메인을 포함할 수 있다.
추가의 실시 양태에서, cCAR은 CAR의 1개 또는 2개 또는 다수의 단위를 포함할 수 있다. 각 단위 CAR은 동일하거나 상이한 힌지 영역 및 보조 자극 도메인을 지닐 수 있다.
추가의 실시 양태에서, 표적 항원은 ROR1, PSMA, MAGE A3, 당지질, 글리피칸 3, F77, GD-2, WT1, CEA, HER-2/neu, MAGE-3, MAGE-4, MAGE-5, MAGE- 6, 알파태아단백, CA-19-9, CA-72-4, NY-ESO, FAP, ErbB, c-Met, MART-1, CD30, EGFRvIII, 면역글로불린 카파 및 람다, CD38, CD52, CD3, CD4, CD8, CD5, CD7, CD2 및 CD138의 그룹 중 적어도 하나를 포함할 수 있으나, 이에 제한되지는 않는다. 표적 항원은 또한 인간 유두종 바이러스 (HPV) 또는 EBV (엡스타인 바 바이러스) 항원으로부터의 E6 및 E7과 같은 바이러스 또는 진균 항원을 포함할 수 있다.
일부 실시 양태에서, cCAR은 CD19 또는 CD20 항원 또는 이들 모두를 발현하는 세포를 표적으로 한다. 또 다른 실시 양태에서, cCAR은 CD19 또는 CD22 항원 또는 이들 모두를 발현하는 세포를 표적으로 한다. 표적 세포는 B 세포 림프종 또는 백혈병과 같은 암세포이다.
급성 이식편-대-숙주 질환 (GVHD)은 동종이계 조혈 줄기 세포 이식 후의 이환율 및 사망률의 가장 중요한 원인으로 남아있다. GVHD의 효과기 단계에서, 알파 및 베타 사슬의 이질이합체인 T 세포 수용체 (TCR)는 T 세포 표면에 발현되며, TCR은 숙주 세포 상 HLA 분자 상의 일부 항원을 인식하고 T 세포 증식을 증진시키고 숙주 세포에 손상을 일으키는 세포 독성제를 방출한다. TCR 유전자 불활성화는 잠재적 이식편-대-숙주 반응을 방지하는데 효율적이다. TCR의 불활성화는 동종항원 및 따라서 GVHD의 TCR 인식의 방지를 야기할 수 있다.
NK 세포 상 CD45의 역할은 T 세포의 그것과 다소 다르다. CD45-결핍 마우스으로부터 유래된 NK 세포는 원형 종양 세포주인 Yac-1에 대해 정상적인 세포독성 활성을 갖는다. 또한, CD45-결핍 NK 세포는 정상적으로 증식하고 IL15 및 IL-21에 반응한다. 따라서, CD45 파괴 또는 제거는 NK 세포의 살상 및 증식에 영향을 주지 않을 것이다. 본 개시내용은 T 또는 NK 세포에서의 CD45를 영구적으로 제거하고, 이어서 CD45-특이적 CAR을 안정적으로 도입하는 방법을 포함한다. 결과적으로, 조작된 T 세포는 항원 제시에 대한 자가-살상 및 반응을 일으키는 것이 없이 CD45에 대한 재지시된 특이성의 목적하는 성질을 나타낸다. 추가의 실시 양태에서, 조작된 T 세포는 악성 종양 또는 다른 질환을 치료하기 위한 기성품 요법으로서의 효능을 가질 수 있다.
본 개시내용은 내인성 CD45의 불활성화 또는 제거를 통해 TCR 신호전달이 감소되거나 손실될 때 증식이 허용되도록 T- 세포가 조작되는 방법에 관한 것이다. TCR 신호전달의 감소 또는 손실은 GVHD를 방지하는 결과를 낳을 수 있다.
추가의 실시 양태에서, CD45의 불활성화에 의해 TCR 신호전달을 감소시키거나 손실 하는 T 세포는 "기성품(off the shelf)"치료 제품으로서 사용될 수 있다.
본 개시내용은 T 또는 NK 세포를 변형하는 방법을 포함하며, 이 방법은 (a) CD45를 불활성화시키는 것에 의해 T 또는 NK 세포를 변형시키는 것; (b) 이들 변형된 세포를 팽창시키는 것; (c) CD45를 발현하지 않는 변형된 T 또는 NK 세포를 분류하는 것; (d) CD45CAR을 도입하는 것을 포함한다.
실시 양태에서, CD45CAR 유전자는 항원 인식 도메인, 힌지 영역, 막통과 도메인 및 T 세포 활성화 도메인 중 적어도 하나를 포함하는 키메라 항원 수용체 (CAR)를 코딩하며, 항원 인식 도메인은 세포 상 존재하는 CD45 표면 항원에 대비해 재지시된다. 항원 인식 도메인은 CD45 항원에 대해 지시된 단클론 항체 또는 다클론 항체를 포함한다. 항원 인식 도메인은 단클론 또는 다클론 항체의 결합 부분 또는 가변 영역을 포함한다.
일부 실시 양태에서, 변형된 T 세포는 동종이계 공여자로부터 얻어지고 '기성품 제품 (off-the-shelf product)'으로서 사용된다.
CAR T 또는 NK 세포를 사용하여 CD45를 표적으로 하는 것은, T 및 NK 세포가 이러한 표면 항원을 발현시키기 때문에 자가 살상을 일으킬 수 있다. 이 결점을 극복하기 위해, 본 발명자는 조작된 CRISPR/Cas9 시스템, 징크핑거뉴클레아제(ZFN) 및 TALE 뉴클레아제(TALEN) 및 메가뉴클레아제를 사용하여 CD45 유전자를 불활성화시키는 것을 제안한다. T 또는 NK 세포에서의 CD45의 손실은 CD45를 발현하는 신생물을 표적으로 하는 CAR에 추가로 형질 도입된다.
본 개시내용은 골수, 혈액 및 기관에서 비정상 세포 또는 악성 세포를 제거 또는 감소시키는 방법을 포함한다. 일부 실시 양태에서, CD45를 발현하는 악성 세포는 급성 백혈병, 만성 백혈병, B 및 T 세포 림프종, 골수성 백혈병, 급성 림프모구성 림프종 또는 백혈병, 원발성 삼출성 림프종, 망상조직세포종, 다운 증후군의 일과성 골수증식성 장애, 림프구우세 호지킨 림프종, 골수성 백혈병 또는 육종, 가지세포종, 조직구성 육종, 건막 거대 세포종, 수지양 수지상세포육종, 이식 후 림프 증식성 장애 등을 가진 환자에서 존재한다.
일부 실시 양태에서, CD45CAR 세포는, 조혈 세포를 제거하는 동시에, 이식 거부 가능한 백혈병/림프종 세포 또는 면역 세포를 제거함으로써 골수에서 골수 줄기 세포 이식을 위한 골수에서 공간을 형성하는데 사용될 수 있다.
추가의 실시 양태에서, CD45CAR 세포는 줄기 세포를 받기 위한 골수 이식을 받기 전에 환자의 예비처리에 사용될 수 있다. 추가의 실시 양태에서, CD45CAR은 조혈 줄기세포 이식을 위한 골수절제 조건화 요법으로서 사용될 수 있다.
일부 실시 양태에서, CD45CAR 세포는 줄기 세포 이식 및/또는 화학요법 후의 잔류 질환을 치료 또는 방지하는데 이용된다.
일부 실시 양태에서, CD45CAR은 발현하는 유전자 또는 카세트의 일부이다. 바람직한 실시 양태에서, 발현하는 유전자 또는 카세트는 CD45CAR 이외에 보조 유전자 또는 태그 또는 이의 일부를 포함할 수 있다. 보조 유전자는 카스파제 9유전자, 티미딘 키나아제, 시토신데아미나제(CD) 또는 시토크롬 P450을 포함하나 이에 한정되지는 않는 유도성 자살 유전자 또는 이의 일부일 수 있다. "자살 유전자" 제거 접근법은 유전자 요법의 안전성을 개선시키고 특정 화합물이나 또는 분자에 의해 활성화되는 경우에만 세포를 살상시킨다. 일부 실시 양태에서, 자살 유전자는 유도성이고 이합체화의 특정 화학적 유도제 (CID)를 사용하여 활성화된다.
일부 실시 양태에서, 안전 스위치는 c-myc 태그, CD20, CD52 (Campath), 말단절단된 EGFR 유전자 (EGFRt) 또는 이의 부분 또는 조합인 보조 태그를 포함할 수 있다. 보조 태그는 비면역원성 선택 도구로서 또는 마커를 추적하기 위해 사용될 수 있다.
일부 실시 양태에서, 안전 스위치는 RSV F 당단백질 A2 균주 (NSELLSLINDMPITNDQKKLMSNN)의 잔기 254-277에 상응하는 24-잔기 펩타이드를 포함할 수 있다.
일부 실시 양태에서, 안전 스위치는 단클론 항-TNFα 약물에 의해 결합된 TNFα의 아미노산 서열을 포함할 수 있다.
본원에 기술된 임의의 조작된 세포의 투여는 CAR 증폭제의 공동 투여로 보충될 수 있다. CAR 증폭제의 예로는 비제한적으로, 더 좋은 치료 결과를 위해 면역 체크포인트 경로를 표적으로 하는 작용제, 콜로니자극인자-1 수용체 (CSF1R)의 억제제와 같은, CAR 활성을 증진시키는 면역 조절 약물이 포함된다. 면역 체크포인트 경로를 표적으로 하는 작용제는 억제성 면역수용체 CTLA-4, PD-1, 및 PD-L1에 결합하여 CTLA-4 및 PD-1 / PD-L1 차단을 초래하는 소분자, 단백질, 또는 항체를 포함한다. 본원에 사용된 바와 같이, 증폭제는 위에서 기술한 바와 같은 인핸서를 포함한다.
본원에 사용된 바와 같이, "환자"는 포유동물을 포함한다. 본원에 언급된 포유동물은 임의의 포유동물일 수 있다. 본원에 사용된 바와 같이, 용어 "포유동물"은 마우스 및 햄스터와 같은 설치목 포유동물, 및 토끼와 같은 토끼목 포유동물을 포함하나 이에 한정되지는 않는 임의의 포유동물을 지칭한다. 포유동물은 고양이과(고양이) 및 개과(개)를 포함하는 식육목(order Carnivora)으로부터 유래한 것일 수 있다. 포유동물은 소과(소) 및 돼지과(돼지)를 포함하는 우제목(order Artiodactyla), 또는 말과(말)를 포함하는 기제목(order Perssodactyla)으로부터 유래한 것일 수 있다. 포유동물은 영장목(order Primates), 세보이드(Ceboids) 목, 또는 시모이드(Simoids) 목 (원숭이)에 속하거나 유인원 목 (order Anthropoids)(인간 및 유인원)에 속할 수 있다. 바람직하게는, 포유동물은 인간이다. 환자는 대상체를 포함한다.
특정 실시 양태에서, 환자는 0-6개월, 6-12개월, 1-5세, 5-10세, 5-12세, 10-15세, 15-20세, 13-19세, 20-25세, 25-30세, 20-65세, 30-35세, 35-40세, 40-45세, 45-50세, 50-55세 55-60세, 60-65세, 65-70세, 70-75세, 75-80세, 80-85세, 85-90세, 90-95세, 또는 95-100세의 인간이다.
본원에 사용된 바와 같이, 조작된 세포의 "유효량" 및 "치료 유효량"이란 용어는 목적하는 치료적 또는 생리학적 효과 또는 결과를 제공하기 위한 충분한 양의 조작된 세포를 의미한다. 이러한, 효과 또는 결과는 세포 질환의 증상 감소 또는 개선을 포함한다. 목적하지 않는 효과, 예를 들어 부작용은 때로는 목적하는 치료 효과와 함께 나타난다; 그러므로, 진료의는 잠재적 위험과 잠재적 이익 사이에 균형을 잡아서 적절한 "유효량"이 무엇인지 결정한다. 요구되는 정확한 양은 환자의 종, 연령 및 일반적인 상태, 투여 방식 등에 따라 환자마다 다를 것이다. 따라서, 정확한 '유효량'을 명시하는 것은 불가능할 수 있다. 그러나, 임의의 개별 케이스에서의 적절한 "유효량"은 상용 실험만을 사용하여 해당 업계 통상의 기술자에 의해 결정될 수 있다. 일반적으로, 조작된 세포는 표적 세포의 증식을 감소시키기에 충분한 양 및 조건으로 투여된다.
암의 치료, 억제 또는 방지를 위한 전달 시스템을 투여한 후에, 숙련된 진료의에게 잘 알려진 다양한 방법으로 치료적 조작된 세포의 효능을 평가할 수 있다. 예를 들어, 치료적 조작된 세포가 암세포 부하를 감소시키거나 암세포 부하의 추가적 증가를 방지하는 것을 관찰함으로써 화학보조제와 함께 전달되는 치료적 조작된 세포가 환자에서 암을 치료 또는 억제하는 데 효과적이라는 것을 해당 업계 통상의 기술자는 이해할 것이다. 암세포 부하는 당 업계에 공지된 방법, 예를 들어, 중합효소 연쇄 반응 분석을 사용하여 특정 암세포 핵산의 존재 또는 혈액 중 특정 암세포 마커의 식별을 검출하는 것, 예를 들어, 항체 검정을 사용하여 대상체 또는 환자로부터의 샘플 (예를 들어, 비제한적으로, 혈액)에서의 마커의 존재를 검출하는 것, 또는 환자에서의 혈액순환중 암세포 항체 수준을 측정하는 것으로 측정될 수 있다.
본원 명세서 전반에 걸쳐, 양은 범위 및 범위의 상한 및 하한에 의해 정의된다. 각 하한은 각 상한과 조합하여 범위를 정의할 수 있다. 하한과 상한은 각각이 별도의 요소로 고려되어야 한다.
본원 명세서 전반에서 "하나의 실시 양태", "일 실시 양태", "하나의 예" 또는 "일례"에 대한 언급은 실시 양태 또는 예와 관련하여 설명된 특정한 특색, 구조 또는 특징이 본 실시 양태들의 적어도 하나의 실시 양태에 포함된다는 것을 의미한다. 따라서, 본원 명세서 전반에 걸쳐 다양한 곳에서 "하나의 실시 양태에서", "일 실시 양태에서", "하나의 예" 또는 "일례"라는 문구의 출현이 반드시 모두 동일한 실시 양태 또는 예를 지칭하는 것은 아니다. 또한, 특정한 특색, 구조 또는 특징은 하나 이상의 실시 양태 또는 예에서 임의의 적합한 조합 및/또는 하위조합으로 조합될 수 있다. 추가로, 본원에 제공되는 도면은 해당 업계 통상의 기술자를 위한 설명의 목적을 위한 것이고 도면은 반드시 일정한 비율로 그려진 것이 아님이 이해된다.
본원에 사용된 바와 같이, 용어 "구성한다", "구성하는", "포함하다", "포함하는", "가지다", "가지고 있는" 또는 임의의 이들의 다른 변형은 비배타적인 포함을 커버하도록 의도된다. 예를 들어, 요소들의 리스트를 포함하는 프로세스, 물품 또는 장치는 반드시 이들 요소에만 한정되는 것은 아니며, 명확하게 열거되지 않거나 이러한 프로세스, 물품 또는 장치에 내재적인 다른 요소를 포함할 수 있다.
또한, 달리 명백하게 언급되지 않는 한, "또는"은 포괄적인 "또는"을 지칭하고 배타적인 "또는"을 지칭하지 않는다. 예를 들어, 조건 A 또는 B는 다음 중 임의의 하나에 의해 만족된다: A가 참이고(또는 존재하고) B가 거짓이고(또는 존재하지 않고), A가 거짓이고(또는 존재하지 않고) B가 참이고(또는 존재하고), 그리고 A와 B가 모두 참이다(또는 존재한다).
또한, 본원에 주어진 임의의 예 또는 예시는 그들과 같이 사용되는 임의의 용어 또는 용어들의 어떠한 구속으로도 간주되지 않거나, 그러한 것으로 제한하는 것으로 간주되지 않거나, 그 정의를 표현하는 것으로 간주되지 않는다. 대신에, 이러한 예들 또는 예시들은 하나의 특정 실시 양태와 관련하여 설명되는 것으로, 그리고 오직 예시적인 것으로 간주되어야 한다. 이들 예들 또는 예시들과 같이 사용되는 임의의 용어 또는 용어들은, 함께 또는 명세서 내의 다른 곳에서 주어지거나 주어지지 않았을 수 있는 다른 실시 양태들을 포괄할 것이고, 이러한 모든 실시 양태들은 해당 용어 또는 용어들의 범위 내에 포함되도록 의도된다는 것임을 해당 업계 통상의 기술자는 이해할 것이다. 그러한 비제한적 예 및 예시를 나타내는 언어는 "예를 들어", "예컨대", "예" 및 "하나의 실시 양태에서"를 포함하나 이에 한정되지는 않는다.
이 명세서에서는 다수의 멤버를 포함하는 다양한 매개 변수 그룹이 기재된다. 매개 변수의 한 그룹 내에서, 각 멤버는 다른 멤버 중 1개 이상과 조합하여 추가 하위 그룹을 만들 수 있다. 예를 들어, 만약에 한 그룹의 멤버가 a, b, c, d 및 e이면, 구체적으로 고려되는 추가 하위 그룹은 멤버 중 임의의 1개, 2개, 3개 또는 4개를 포함하며, 예를 들어, a 및 c; a, d 및 e; b, c, d 및 e; 등을 포함한다.
본원에 사용된 바와 같이, XXXX 항원 인식 도메인은 XXXX에 대해 선택적인 폴리펩타이드이다. 따라서 XXXX가 표적이다. 예를 들어, CD38 항원 인식 도메인은 CD38에 특이적인 폴리펩타이드이다.
본원에 사용된 바와 같이, CDXCAR은 CDX 항원 인식 도메인을 갖는 키메라 항원 수용체를 가리킨다.
본 개시내용은 아래에 제시되는 예를 참조하여 더 잘 이해될 수 있다. 다음의 예는 본원에 청구된 화합물, 조성물, 제품, 장치 및/또는 방법이 어떻게 제조되고 평가되는지에 대한 완전한 개시 및 설명을 해당 업계 통상의 기술자에게 제공하기 위해 제시되며, 순수하게 예시적인 것으로 의도되며, 본 개시내용을 제한하려는 것은 아니다.
실시예
화합물 CAR (
cCAR
)의 생성
CD33CD123 cCAR의 구축물은 도 1A의 도식을 따른다. 여기에는 2개의 상이한 CAR 단위를 갖는 기능성 화합물 CAR (cCAR)의 발현을 유도하는 SFFV (비장 포커스 형성 바이러스, spleen focus-forming virus) 프로모터가 포함된다. 항원 수용체 맨 윗부분은 항-CD33 및 항-CD123의 scFv (단일-사슬 가변 단편) 뉴클레오타이드 서열이다. 피코르나바이러스로부터 유래된 P2A 펩타이드는 바이시스트론성 (bicistronic) 유전자 구축물에 대한 그의 자가-절단 역학의 고 효율적인 메카니즘 때문에 이용된다. 자가-절단 P2A 펩타이드는 발현동안 CAR, CD33CAR, 및 CD123CAR의 2개의 독립적인 단위를 함께 연결시키는 역할을 한다. 문헌에서 통상적으로 사용되는 내부 리보솜 진입 부위 (IRES)에 대한 이 접근법의 장점은 2A 펩타이드의 상류와 하류의 두 단위 단백질 사이에서의 작은 크기 및 고 절단 효율을 포함한다. 또한, 자가 절단 P2A 펩타이드의 사용은 IRES가 적용될 때 IRES 전후의 유전자 사이의 발현 수준의 차이의 문제를 피할 수 있다.
모듈 단위인 CD33CAR은 CD33 scFv 도메인, CD8a 힌지 영역, CD8a 막통과 도메인, 4-BB 보조-자극 도메인 및 CD3 제타 사슬의 세포내 도메인을 포함한다. 제2 모듈 CAR인, CD123CAR은 CD33CAR과 같은 힌지, 막통과 및 세포내 신호전달 도메인을 가지고 있지만 상이한 scFv 및 보조 자극 도메인을 가지고 있다. CD33 CAR는 그에 상응하는 항원을 인식하고 CD123 CAR는 그에 상응하는 항원과 결합한다. 힌지 영역을 디술피드 상호 작용을 피하는 서열이 되도록 설계하였다. 상이한 보조 자극 도메인, 4-BB 및 CD28을 사용하였다. CD33CD123 화합물 CAR을 렌티바이러스 플라스미드로 서브클로닝하였다.
고효율 화합물 CAR (
cCAR
)의 생성
화합물 CAR 렌티바이러스를, 도 2에 도시된 바와 같이 역가를 증가시키기 위해, 삽입하기에 큰 사이즈로 인해 2배의 벡터 DNA를 제외하고는, 제조업체의 지시에 따라 리포펙타민(Lipofectamine) 2000으로 HEK-293 FT 세포를 형질 감염시킴으로써 생성하였다. 약 12-16시간의 인큐베이션 후에, 리포펙타민을 함유하는 배지를 제거하고 10% FBS, 20mM HEPES, 1mM 피루브산 나트륨 및 1mM 부티르산 나트륨을 함유하는 DMEM으로 교체하였다. 약 24시간 후, 상청액을 채취하고 냉장시키고, 신선한 배지로 교체하였다. 또 다른 약 24 시간 후에, 이를 수집하여 이전의 상청액과 혼합하고 0.45 μM 필터 디스크를 통해 여과하였다. 상청액을 분취량으로 나누어 액체질소로 급속 냉동시키고 -80℃에서 보관하였다. HEK-293 FT 세포를 채취하고, 냉동 보관하고, 후속의 전기영동 및 웨스턴 블롯을 위해 용해시켰다.
PB (말초 혈액) 또는 CB (인간 제대혈) 백혈구연층 세포를 항-CD3 항체 및 IL-2로 2일간 활성화시켰다. cCAR 렌티바이러스 상청액을 레트로넥틴(RetroNectin)으로 코팅된 멀티웰 플레이트(multiwell plate) 위에 스피노큘레이션(spinoculation)하였다. 활성화된 T 세포는 형질도입 효율을 증가시키기 위해 약 0.3 x 106 세포/mL의 저농도에서 렌티바이러스 상청액으로 다수의 웰에서 형질 도입시켰다 (도 2).
첫 번째의 밤샘 형질 도입에 이어, 세포가 건강하지 않게 보이지 않는 한, 세척 없이 두 번째 형질도입을 위해 제2 바이러스-코팅된 플레이트에 세포를 직접 첨가하였다. 두 번째의 밤샘 형질도입에 이어, 세포를 세척하고, 조합하고, 조직 배양 처리된 플레이트에서 인큐베이션하였다. CAR T 세포를 공동 배양 살상 분석 전에 최대 약 5일간 팽창하도록 허용하였다. 약 3일간의 인큐베이션 후, 세포를 바이오틴과 접합된 염소 항-마우스 F(Ab')2 또는 염소 IgG (동형) 항체와 함께 인큐베이션하고, 세척하고, 이어서 스트렙타비딘-PE (streptavidin-PE) 및 접합된 항-인간 CD3와 함께 인큐베이션하였다. 세포를 세척하고 2% 포르말린에 현탁한 후, 이어서 유동 세포측정법으로 분석하여 형질도입 효율의 백분율을 결정하였다.
CD33CD123
cCAR의
특징화
화합물 구축물을 확인하기 위해 형질 감염된 CD33CD123 cCAR HEK293T 세포를 웨스턴 블롯으로 분석하였다. 항-CD3ζ 단클론 항체를 갖는 면역블롯은 화합물 CAR CD3ζ 융합 단백질에 대한 예측된 크기의 밴드를 나타내었다 (도 1B). 중요하게, 비슷한 강도의 두 개의 별개의 밴드가 블롯 상에서 관찰되었으며, 이는 예상한 바와 같은 P2A 펩타이드의 성공적인 고 절단 작용을 시사한다. 예상한 대로 GFP 대조 벡터에 대해 CD3ζ 발현은 나타나지 않았다. scFv의 표면 발현을 또한 HEK 293 세포 (도 1C) 및 1차 T 세포 (도 1C) 상에서 시험하였다.
화합물 CD33CD23CAR 렌티바이러스를 HEK293 세포주에서 형질 도입 효율에 대해 시험하고 유동 세포측정법으로 분석하였다 (Beckman Coulter) (도 1C). 유동 세포측정법은 HEK 세포의 약 67%가 CD33CD123 CAR를 발현한다는 것을 보여 주었다. 인간 말초 혈액 (PB)은 자가 T 세포 요법에 자주 사용된다. 인간 PB 백혈구 연층 세포를 항-CD3 항체와 IL-2로 활성화하고, CD4CAR 또는 대조 (GFP) 렌티바이러스로 형질도입시켰다. 형질 도입 후, 유동 세포측정법 분석은 T 세포의 약 22%가 CD33CD123CAR을 발현한다는 것을 보여 주었다 (도 1C).
결과
제대혈(
UCB
) 및 말초 혈액(PB)에서 유래한
CD33CD123
cCAR
T 세포는 CD33-발현 종양 세포를 특이적으로
살상한다
CD33CD123 cCAR T 세포 또는 GFP T 세포 (대조)를 표적 세포와 0.5:1 내지 50: 1, 바람직하게는 약 2:1, 5:1, 10:1, 20:1, 50:1의 범위의 비율로, 각각 약 100,000, 200,000, 500,000, 약 1백만, 또는 2백만 개의 효과기 세포 대 약 50,000, 100,000, 200,000 개의 표적 세포로, 약 1-2 mL T 세포 배양 배지에서, IL-2 없이 약 24시간 동안 인큐베이션하였다. 표적 세포는 백혈병 환자로부터의 백혈병 세포주 및 백혈병 세포였다. 약 24시간의 공동 배양 후, 세포를 마우스 항-인간 CD33, CD123, CD34 및 CD3 항체로 염색하였다.
CD33 CAR 및 CD123 CAR를 발현하는 CD33CD123 cCART 세포를 생성하고 HL60 및 KG-1a 세포주를 사용하여 항-백혈병 기능에 대해 시험하였다. HL60 세포주는 CD33이 매우 풍부한 전골수구성세포백혈병 세포주이다. 이의 세포 집단의 약 100%가 CD33+이고 이의 작은 하위세트(<10%)가 희미한 CD123+이다. 배양에서, CD33CD123 CAR의 유효성을 측정하기 위해 CD33-발현 백혈병 세포를 표적화하는 것에 중점을 두고 이 세포주를 시험하였다. 또한, HL60에서의 CD33의 강력한 발현때문에, CD33CD123 cCAR 효과는 엄청날 수 있다. 실제로, 다양한 비율의 효과기 세포 대 표적 세포를 포함하는, 24시간의 공동 배양 조건에서, CD33CD123 cCAR은 유의한 백혈병 세포 살상 특성을 나타내었다 (도 4). CB-유래 CD33CD123 CAR T 세포를 HL60 세포를 살상하는 그의 능력에 대해 먼저 시험하였다. 약 24 시간 인큐베이션 및 약 0.5:1 내지 50:1, 바람직하게는 1:1 내지 약 5:1, 더 바람직하게는 약 2:1 내지 4:1 범위의 낮은 효과기 대 표적 (E:T) 비율에서, CD33CD123 CAR 세포는 GFP 대조와 비교할 때 약 55%의 CD33 발현 HL60 세포를 제거하였다. 약 5:1의 비율에서, 살상 작용은 약 82%까지 상승하였다.
골수성 백혈병 세포주 KG1a와 말초 혈액 단핵 세포 (PBMCs)로부터 유래된 CD33CD123 CAR를 공동 배양하였는데, 이는 또한 HL60 및 50-80% CD123과 비교하여 적정한 수준에서 약 100% CD33을 발현하였다. 따라서 KG1a는 CD33CD123 CAR가 표적으로 하는 항원에 대해 이중 양성인, 상대적으로 이중적인 표적 세포 집단이다. 약 24시간의 인큐베이션 및 약 0.5:1 내지 50:1 범위의 낮은 효과기:표적 (E:T) 비율을 사용하였다. 약 2:1의 낮은 E:T 비율에도 불구하고, CD33CD123 CAR은 약 26%의 보통의 항백혈병 활성을 나타내었고, E:T 비율을 10:1까지 증가시켰을 때 GFP 대조와 비교하여 약 62%의 KG1a 살상을 초래하였으며 (도 5), 이는 KG1a보다 강하게 CAR 작용을 나타내고 이를 이용하는 HL60을 사용하면 CD33 마커의 강도가 살상의 효능에 대한 지표가 될 수 있다는 것을 시사한다. 이러한 실험은 관련 항원 제시 세포 집단에 대한 전체 CD33CD123 CAR의 기능에 대한 증거를 제공한다.
추가 화합물 CAR, CD33CD123-BB cCAR을 생성하였다. 이 화합물 CAR은 CAR의 2개의 독립 단위, CD33 및 CD123을 포함한다. 제1 CAR은 CD33에 결합하는 scFv를 포함하고, 제2 CAR은 CD123을 인식하는 상이한 scFv를 갖는다. 두 CAR 모두 동일한 힌지 영역, 막통과, 보조 자극 및 세포내 도메인을 포함한다. CD33CD123-BB cCAR 렌티바이러스를 생산하고 그들의 살상 능력을 KG-1a 세포에서 시험하였다. 도 5에 도시된 바와 같이, 약 10:1의 비율에서 실질적인 살상이 있었지만, 그것은 CD33CD123 cCAR의 것 보다 덜 강력하다.
CD33CD123
cCAR은
CD33 및/또는 CD123을 발현하는 환자 샘플에 대하여 활성을 가지고
있다
세포주 실험 이외에, 각 개개의 CAR 단위의 기능을 시험하기 위해 환자 샘플에 대한 연구도 수행하였다. 공격성 급성 골수성 백혈병(AML)인 AML-9를 CD33CD123 cCAR의 효능을 시험하는 데 사용하였다. AML-9 샘플에서의 다수의 세포 유형을 포함하는 환자 세포 집단의 이질성 때문에, 백혈병 모세포를 CD34 및 CD33으로 게이팅하였는데, 이는 백혈병 모세포가 이들 두 마커에 대해 양성이었기 때문이다. 이 백혈병 세포의 CD33+ CD34+ 집단의 고갈은 CAR T 세포: 표적 세포의 비율에서 GFP 대조와 비교했을 때 48%인 것으로 관찰되었다 (도 6).
CD123 양성 및 CD33 음성인 백혈병 세포도 시험하였다. 이 목적을 위해 인간 B 세포 급성 림프모구성 백혈병 (B-ALL) 샘플인 Sp-BM-B6을 선택했다. 이 샘플의 모든 백혈병 모세포는 CD34+ CD33-이고, CD123에 대해 약 50% 초과 양성이었다. CD33CD123 cCAR T 세포에 의한 CD34+ 백혈병 세포 집단의 고갈은 GFP 대조의 것과 비교했을 때 약 86%였다(도 7). 세포주 및 인간 샘플 연구에 기반하여, 우리의 데이터는 화합물 CD33CD123 CAR이 CD33 또는 CD123 또는 이들 모두를 발현하는 백혈병 세포를 표적화할 수 있음을 강력히 시사한다.
CD33 또는 CD123 또는 이들 모두를 발현하는 백혈병 세포를 표적으로 하는 CD33CD123
cCAR
NK
세포
자연 살상 (NK) 세포는 CD56+CD3-이며 CD8+ T 세포처럼 감염된 및 종양 세포를 효과적으로 살상할 수 있다. CD8+ T 세포와는 달리, NK 세포는 세포를 살상하는 것에 대한 활성화 요구 없이 종양에 대해 세포 독성을 개시한다. NK 세포는 사이토카인 폭풍의 잠재적으로 치명적인 합병증을 피할 수 있기 때문에, 보다 안전한 효과기 세포이다. 그러나, 백혈병을 살상하는 데 CD33 또는 CD123 또는 둘 다를 사용하는 것은 완전히 연구되지 않았다.
CD33CD123
cCAR
NK
세포의 생산
2번의 연속적 밤샘 형질 도입으로 NK-92 세포를 CD33CD123 CAR 렌티바이러스 상청액으로 형질 도입시키고 중간에 레트로넥틴 (retronectin) 및 바이러스가 코팅된 플레이트로 바꾸었다. 형질 도입된 세포를 3일 또는 4일 동안 팽창시킨 후, CAR 발현을 위해 유동 세포측정법으로 분석하였다. 세포를 채취하고 염소 항-마우스 F(Ab')2와 함께 약 1:250으로 약 30분 동안 인큐베이션하였다. 세포를 세척하고, 현탁시키고, 스트렙타비딘-PE(streptavidin-PE )로 약 30분 동안 염색하였다. 세포를 세척하고 2% 포르말린에 현탁시키고, 유동 세포측정법으로 분석하였다. 이어서, CD33CD123 cCAR을 발현하는 NK-92 세포를 상기와 같이 표지하고, FACSAria상에서 분류하여 F(Ab')2-발현 세포의 상위 0.2%를 수집하고 배양하였다. 분류되고 팽창된 세포의 후속 표지는 NK-92 세포의 약 89%가 항-마우스 F(Ab')2에 대해 양성임을 나타냈다 (도 8).
CD33CD123
cCAR
NK
세포는 백혈병 세포를 효율적으로 용해하거나
제거한다
우선, 공동 배양에서의 HL-60 암 세포주를 살상하는 CD33CD123 cCAR NK-92 세포의 능력을 평가함으로써 CD33CD123 cCAR NK-92 세포의 기능을 시험하였다. 사실상 모든 HL-60 세포는 CD33을 고도로 발현하지만 이 세포주에서의 CD123 발현은 단지 10% (약한) 미만이다. 그러므로, CD33CD123cCAR의 살상 능력은 CD33을 적절하게 표적화할 수 있는 cCAR의 능력에 의존할 가능성이 높다.
CD33CD123 cCAR NK-92 세포를 IL-2 없이 NK 세포 배지에서 약 24시간 동안 HL-60 세포와 공동 배양하였다. 인큐베이션 후, CD33CD123 cCAR NK-92 세포를 표지하고, 비-CAR, GFP NK-92 세포의 대조와 비교하였다. CD33CD123 cCAR NK-92 세포에 의한 HL-60 세포의 극적인 살상을 대조, GFP NK-92 세포와 비교하여 관찰하였다. 더욱이, CD33CD123 cCAR NK-92 세포의 살상 능력은 대조와 비교하여 약 10대1 비율에서 약 100%로 용량-의존적이었다 (도 9 및 도 11).
골수성 백혈병 세포주를 이용한 두 번째 공동 배양 실험을, 모든 세포에서 CD33을 발현하는 KG1a를 사용하여 수행하였지만, HL-60의 것에 비해 보통 수준이었다. CD123 항원은 KG1a 세포의 약 50-80%에서 발현된다. 실험 디자인은 상기 기재된 HL-60 살상 분석의 첫 번째 실험과 유사하게 동일한 인큐베이션 시간, 효과기:암세포 비율 및 GFP NK-92 세포 대조를 사용하였다. 결과는 GFP NK-92 세포 대조와 비교할 때 용량-의존적 방식의 CD33CD123 cCAR NK-92 세포에 의한 현저한 KG1a 세포 살상을 보여준다. 10:1 효과기:표적의 비율에서, CD33CD123 cCAR NK-92 세포에 의한 KG1a 세포 살상은 GFP 대조의 것과 비교하여 약 85%였다 (도 10 및 11).
KG1a 세포의 분석은 2개의 상이한 집단, CD33+CD123- 및 CD33+CD123+을 보여주었다. 도 12은 세포 살상에 있어서 두 집단 모두에서 나타난 용량-의존적 증가를 나타낸다. 놀랍게도, 이중 양성 집단은 각 증가된 비율에 대해 더 높은 효율적인 살상을 나타냈고, 이는 CD33 및 CD123의 두 개의 모듈 CAR의 가능한 상승작용적 효과를 시사한다 (도 12).
CD19CD20
,
CD19CD22
,
CD19CD138
cAR의
생성
위에서 설명된 CD33CD123 cCAR의 것과 유사한 전략을 사용하여 3개의 Ccar을 생성하였다 (도 13).
다발성 골수종 치료를 위한
BCMA
CS1
cCAR
및
BCMA
CD19
cCAR를
포함하는 cCAR의 생성
CD38, CS1, CD138, B 세포 성숙 항원 (BCMA) 및 CD38을 포함하는 표면 항원을 표적화하기 위한 cCAR에 대한 전임상 연구를 진행하였다. CD19 CAR은 1상 임상 시험에서 다발성 골수종 치료에 약간의 효능을 나타냈다. 그러나 표적 항원 발현의 이질성이 악성 형질 세포에서 통상적으로 발생한다는 점을 고려하면 (Ruiz-Arguelles and San Miguel 1994), 단일 표적이 이 질환을 제거하기에 충분할 가능성은 낮다. BCMA CS1 cCAR, BCMA CD19 cCAR, BCMA CD38 cCAR 및 BCMA CD138 cCAR을 생성하였고, 실험 디자인은 위에서 설명된 바와 같이 CD33CD123 cCAR의 것과 유사하였다.
다발성 골수종 치료를 위한
BCMA
CS1
cCAR
(
BC1cCAR
)을 포함하는
cCAR
생성
BCMA
-CS1
cCAR
(
BC1cCAR
) 구축물의 생성 및
특징화
BC1cCAR의 모듈 디자인은 자가 절단 P2A 펩타이드에 의해 항-CD319 (CS1) scFv에 융합된 항-CD269 (BCMA, B 세포 성숙 항원) 단일사슬 가변 단편 (scFv) 영역, CD8-유래된 힌지(H) 및 막통과(TM) 영역, 및 CD3ζ 신호전달 도메인에 연결된 직열(tandem) 4-1BB 공동-활성화 도메인으로 구성된다 (도 14A). 강력한 비장 포커스 형성 바이러스 프로모터 (SFFV) 및 CD8 리더 서열을 T 세포 표면 상에서의 BC1cCAR 분자의 효율적인 발현을 위해 사용하였다. 두 단위의 CAR은 같은 보조 자극 도메인, 4-1BB를 사용하였다. 형질 감염된 BC1cCAR HEK293T 세포를 화합물 구축물을 확인하기 위해 웨스턴 블롯 분석에 적용하였다. 항-CD3ζ 단클론 항체를 사용한 면역블롯은 화합물 CAR CD3ζ 융합 단백질에 대한 예측된 크기의 밴드를 나타내었다 (도 14E). 중요하게, 비슷한 강도의 2개의 별개의 밴드가 블롯 상 관찰되었으며, 이는 예상한 바와 같은 P2A 펩타이드의 성공적인 고 절단 작용을 시사한다. 예상한대로 GFP 대조 벡터에 대해 CD3ζ 발현은 나타나지 않았다.
BC1cCAR
(
cCAR
) T 세포의 생성
제대혈 (UCB) 백혈구연층으로부터 단리된 T 세포를 2일의 활성화 후에 BC1cCAR 렌티바이러스로 형질도입시켰다. 두 단위의 CAR은 같은 보조 자극 도메인, 4-1BB를 사용했다. BC1cCAR의 형질 도입 효율은 유동 세포측정법에 의해 측정시 약 15%로 결정되었다 (도 14B). BC1cCAR T 세포를 먼저 골수종 마커, BCMA 및 CS1에 대해 음성인 CML (만성 골수성 백혈병) 세포주에서 시험했다. 예상한 바와 같이, 야생형 K562에 대한 대조 T-세포 또는 BC1cCAR T-세포로부터 용해는 일어나지 않았다 (도 14C). BCMA-K562 (Kochenderfer, NIH)는 80% 이상의 세포 집단에서 BCMA를 발현하게 하려고 BCMA 발현 cDNA로 형질 도입된 K562 세포였다. BC1cCAR T 세포를 이 세포주와 2:1 및 5:1의 E:T 비율에서 공동 배양하여, 대조 (검출 불가능)와 비교했을 때 30% 이상의 용해를 나타냈다 (도 14C). 이런 결과는 CS1CAR T-세포와 같은 다른 CAR에 대해 항원-형질 도입된 세포주에서 수행된 다른 배양과 부합한다.
그러나, BCMA-CS1-2G (cCAR 일종)가 각 단위에 대해 상이한 보조 자극 도메인, 4-BB 또는 CD28을 사용한 경우, 희귀한 표면 CAR 발현이 검출되었는데, 이는 보조 자극 도메인의 적절한 선택이 T 세포 상에서의 표면 CAR 발현을 보장하는데 중요하다는 것을 나타낸다(도 14D). 비록 단백질을 HEK 세포에서 웨스턴 블롯팅에 의해 검출했지만 (도 14E), CD269-CS1-2G 렌티바이러스 상청액으로 형질 도입된 활성화된 T 세포에서는 표면 발현을 검출할 수 없었다. 이것은 발현된 단백질을 세포막으로 내보낼 수 없기 때문일 수 있다. 앞으로, 우리는 더욱 큰 세포 표면 발현을 가능하게 하기 위해 이 구축물의 서열을 최적화할 필요가 있을 수 있다.
BC1cCAR
T 세포는
BCMA
+
및 CS1
+
세포주를 특이적으로
용해시킨다
BC1cCAR T 세포의 세포 독성 능력을 평가하기 위해, MM1S (BMCA+ CS1+), RPMI-8226 (BCMA+ CS1-), 및 U266 (BCMA+ CS1dim)의 골수종 세포주와의 공동 배양 분석을 수행했다. 표적 세포를 용해시키는 BC1cCAR T 세포의 능력을 유동 세포 측정법 분석으로 정량화하였고 표적 세포를 사이토트래커(Cytotracker) 염료 (CMTMR)로 염색하였다. 24시간의 공동 배양에서, 2:1의 E:T 비율에서의 표적 세포의 90% 이상 고갈 및 5:1의 E:T에서의 95% 이상 고갈로 BC1cCAR은 MM1S 세포의 사실상 완전한 용해를 나타냈다 (도 15). RPMI-8226 세포에서, BC1cCAR은 2:1의 E:T 비율에서 70% 이상의 BCMA+ 표적 세포 및 5:1의 E:T에서 75% 이상 용해시켰다 (도 16). U266 표적 세포와의 24시간 공동 배양에서, BC1cCAR은 E:T 비율 2:1에서 BCMA+ U266 세포 80%를 용해해 포화 상태에 도달하였다 (도 17).
BC1cCAR
T 세포는 원발성 환자 골수종 샘플에서
BCMA
+
및 CS1
+
집단을 특이적으로
표적화한다
MM10-G 환자 샘플의 유동 세포측정법 분석은 뚜렷하고 일관된 BCMA+ 및 CS1+ 집단 하위세트를 보여준다 (도 18). MM7-G 샘플은 완전한 BCMA+ CS1+ 표현형을 나타내지만, MM11-G는 아마도 골수 흡인물인 그의 특성에 기인하는 노이즈 BCMAdim CS1dim 표현형을 보여준다. 24시간 후, BC1cCAR T 세포는 5:1의 E:T 비율에서 75% 이상인 MM7-G 원발성 환자 샘플의 강한 제거를 나타내고, 10:1에서는 85% 이상으로 증가한다 (도 19). MM11-G (도 20)에 대하여, BC1cCAR T 세포는 10:1의 E:T에서 BCMA+CS1+ 집단의 45% 이상을 용해시킬 수 있었다.
BC1cCAR은 24시간에 걸쳐 MM10-G 공동 배양에서 BCMA+CS1+ 및 BCMA-CS1+ 집단 하위세트 둘 모두를 유의하게 제거함으로써 표적화된 및 특이적 용해 능력을 보여준다. 2:1의 E:T 비율에서, BC1cCAR T 세포는 BCMA+CS1+ 집단의 60% 이상 및 오직 CS1+뿐인 집단의 70%를 제거한다. 5:1의 E:T 비율에서, 오직 CS1+뿐인 집단의 제거는 80%로 증가한다 (도 18).
BC1cCAR
T 세포는
생체내에서
종양의
유의적인
억제 및 감소를
나타낸다
BC1cCAR T 세포의 생체내 항종양 활성을 평가하기 위해, NSG 마우스를 사용하여 측정 가능한 종양 형성을 유도하기 위해, 치사량에 가깝게 조사하고 다발성 골수종 세포주인, 루시페레이스-발현 MM1S 세포를 정맥내 주사하여 이종 마우스 모델을 개발하였다. 종양 세포 주입 3일 후, 마우스에 8x106 BC1cCAR T 세포 또는 벡터 대조 세포를 1회 용량으로 정맥내 주사하였다. 3, 6, 및 8일째에 마우스에 RediJect D-Luciferin (Perkin Elmer)을 피하 주사하고 종양의 부하를 측정하려고 IVIS 영상을 시행하였다 (도 21). 대조 마우스에 대한 처리된 마우스에서의 종양 세포의 백분율을 측정하기 위해, BC1cCAR T 세포를 주사한 마우스에 대해 측정한 평균 광 강도를 벡터 대조 주입 마우스의 것과 비교하였다 (도 21 및 22). 독립표본 T 검정 분석은 대조와 비교시 BC1cCAR T 세포를 주입한 그룹에서 8일까지 더 적은 광 강도, 따라서 더 적은 종양 부하를 가지는 것으로 두 그룹 간에 극도로 유의한 차이 (P = 0.0001)를 보여주었다 (p <0.0001). 1일째, 그리고 후에 격일마다, 종양 크기 범위를 측정하고 두 그룹 간의 평균 종양 크기를 비교하였다 (도 21). 요약하면, 이런 생체내 데이터는 벡터 대조 NK 대조 세포와 비교할 때 CD269-CS1-BBCAR T 세포가 MM1S 주입된 NSG 마우스에서 종양 부하를 유의하게 감소시킨다는 것을 나타낸다.
CD45 CAR 요법
3쌍의 sgRNA를 관심 유전자를 표적으로 하도록 CHOPCHOP로 디자인한다. 유전자-특이적 sgRNA는 이어서 E2A 자가 절단 링커와 연결된 인간 Cas9 및 퓨로마이신 내성 유전자를 발현하는 렌티바이러스 벡터 (Lenti U6-sgRNA-SFFV-Cas9-puro-wpre)로 클로닝하였다. U6-sgRNA 카세트는 Cas9 요소 앞에 있다. sgRNA 및 Cas9puro의 발현은 각각 U6 프로모터 및 SFFV 프로모터에 의해 유도된다 (도 23).
이하의 유전자-특이적 sgRNA 서열을 사용하고 구축했다.
본 발명의 비제한적인 실시 양태에서, 예시적인 유전자-특이적 sgRNA를 아래에 설명된 바와 같이 디자인하고 구축하였다:
CD45 sgRNA 구축물::
Lenti-U6-sgCD45a-SFFV-Cas9-puro GTGGTGTGAGTAGGTAA
Lenti-U6-sgCD45b-SFFV-Cas9-puro GAGTTTTGCATTGGCGG
Lenti-U6-sgCD45c-SFFV-Cas9-puro GAGGGTGGTTGTCAATG
도 24는 혈액 악성 종양을 표적으로 하는 CD45 CAR T 또는 NK 세포의 생성 단계를 보여준다.
NK
세포 상 CD45를 표적으로 하는
CRISPR
/
Cas
뉴클레아제
유전자-특이적 sgRNA를 운반하는 렌티바이러스를 사용하여 NK-92 세포를 형질 도입시켰다. NK-92 세포 상에서의 CD45 발현의 손실은 유동 세포측정법 분석에 의해 측정하였다. NK-92 세포의 CD45 음성 집단을 분류하고 팽창시켰다 (도 25). 분류 및 팽창된 CD45 음성 NK-92 세포를 사용하여 CD45CAR NK 세포를 생성하였다. 얻어진 CD45CAR NK 세포를 사용하여 CD45+ 세포를 살상하는 그의 능력을 시험하였다.
CRISPR
/
Cas
뉴클레아제 표적 후 CD45 불활성화
NK
-92 세포 (
NK
45i
-
92)의
기능적
특징화
CD45의 CRISPR/Cas 뉴클레아제 불활성화 후, NK45i-92 세포의 성장이 야생 NK-92 세포의 것과 유사하다는 것을 입증하였다 (도 26). CD45의 불활성화는 NK-92의 세포 증식에 유의한 영향을 미치지 않았다. 또한, 세포를 백혈병 세포인 CCRF와 공동 배양했을 때 NK45i-92 세포의 용해 능력이 야생형 NK-92의 것과 양립할 수 있다는 것을 밝혔다 (도 27).
CD45-불활성화된 NK-92가 CAR 용해와 양립할 수 있었다는 것을 입증하기 위해, NK45i-92 세포 및 그들의 야생형 NK-92를 CD5CAR 또는 GFP를 발현하는 렌티바이러스로 형질 도입시켰다. 얻어진 CD5CAR NK45i-92 세포 및 GFP NK45i-92를 FACS로 분류하여 표적화 세포를 살상하는 그의 능력을 비교하는 데 사용했다. CD5CAR NK45i-92 세포는 CCRF-CEM 세포와 공동 배양하였을 때, 비율 (E:T), 2:1 및 5:1에서 CD5 표적 백혈병 세포를 강하게 살상하는 능력을 보여주었다. CD5CAR NK45i-92와 CD5 CAR NK-92 세포 사이에 시험관내 CCRF-CEM 세포의 제거의 유사한 효능이 있는 것으로 나타났다 (도 28). 이것은 CD45 발현의 손실이 CAR NK-세포의 항 종양 활성을 감소시키지 않는다는 것을 시사한다.
CD45CAR
구축물의 생성
다음으로 NK45i-92 세포에서의 CD45CAR이 백혈병 세포에서의 CD45 항원에 반응하는 것을 조사한다. 우리는 CD45CAR을 생성했다. CD45CAR은 항-CD45 단일 사슬 가변 단편 (scFv) 영역, CD8-유래 힌지 (H) 및 막통과 (TM) 영역, 및 CD3ζ 신호전달 도메인에 연결된 직열 CD28 및 4-1BB 공동-활성화 도메인으로 구성된다 (도 29A). 강력한 비장 포커스 형성 바이러스 프로모터 (SFFV) 및 CD8 리더 서열을 사용하였다. CD45CAR 단백질을 적절한 벡터 대조를 가진 CD45CAR 렌티바이러스 플라스미드로 형질감염된 HEK293-FT 세포의 웨스턴 블롯에 의해 특징화하였다. 추가적으로, 항-CD3제타 단클론 항체 면역 블롯은 CD45CAR 단백질에 대한 예측된 크기의 밴드를 나타내었고 벡터 대조에서는 밴드가 관찰되지 않았다 (도 29B).
CD45CAR
NK
45i
-92
NK
세포
NK45i-92 세포를 풍부화하기 위한 형광 활성화된 세포 분류 (FACS) 후, CD45CAR NK-92 형질 도입 효율은 분류 후의 유동 세포측정법에 의해 결정된 바와 같이 87%인 것으로 결정되었다 (도 30). NK45i-92 세포의 FACS 수집 후, CD45CAR 발현 수준은 적어도 10 회의 계대 동안 일관되게 안정적으로 유지되었다.
CD45CAR
NK
45i
-92 세포는 CD45+ 백혈병 세포를 특이적으로
용해시킨다
CD45CAR NK45i-92 항-백혈병 활성을 평가하기 위해, T-ALL 세포주, CCRF-CEM 및 Jurkat, 및 NK 세포주 및 NK-92 세포를, 이들 모두가 CD45를 발현하기 때문에, 사용하여 공동 배양 분석을 수행하였다 (도 31, 32 및 33). CD45CAR NK45i-92 세포가 백혈병 세포의 강력한 용해를 일관되게 나타냈다는 것을 입증했다. 낮은 효과기 대 표적 세포 (E:T 비율 5:1)에서의 6시간 인큐베이션 후, CD45CAR NK45i-92 세포는 60% 초과의 CCRF-CEM 세포를 효과적으로 용해시켰다 (도 31). 6 시간 공동 배양 후, CD45CAR NK45i-92 세포는 또한 2:1 또는 5:1의 E:T 비율에서 약 60% Jurkat 세포를 제거할 수 있었다 (도 32). 6 시간의 공동 배양 후, CD45CAR NK45i-92 세포는 2:1의 E:T 비율에서 20% CD45 양성 NK-92 세포를 효율적으로 용해시켰고 5:1의 E:T 비율에서 60% 가까이 용해시켰다 (도 33A-33C).
혈액 악성 종양에 대한 CD45 표적을 추가로 분석하기 위해, 또한 추가의 두 개 CAR: CD45-28 및 CD45-BB를 생성하였고, 그리고 CD45-28 또는 CD45-BB CAR를 발현하는 렌티바이러스를 사용하여 NK45i-92 세포를 형질 도입시켰다. CD45-28 및 CD45-BB CAR에는 위에서 설명한 CD45CAR의 그것과 다른 새로운 항-CD45 scFv가 포함되어 있다. CD45-28 CAR은 CD28 보조 자극 도메인을 사용하지만 CD45-BB는 4-BB 보조 자극 도메인을 가지고 있다. 두 CAR 모두 CD8 유래 힌지 (H), 막통과 (TM) 영역 및 CD3ζ 신호전달 도메인을 사용한다. CD45CAR은 B 급성 림프모구성 세포주, REH의 강력한 용해를 나타낸다. CD45CAR NK45i-92 세포는 약 76%의 REH 세포를 용해시켰다. CD45b-BB CAR NK45i-92 세포 및 CD45b-28 CAR NK45i-92 세포는 대조 GFP NK-92 세포와 비교하여 각각 REH 세포의 약 79% 및 100% 용해를 나타냈다 (도 33D-G). CD45b-28 CAR NK45i-92 세포는 REH 세포의 최고의 용해 능력을 나타냈다.
CAR T 및
NK
세포 기능을 증진시키는 데 있어서의 IL15 및 이의 수용체
최근 연구는 T 세포의 지속성이 CAR T 세포 치료 효능과 잘 연관되어 있음을 입증하였다. 최근의 시험은 주입된 소량의 CAR T 세포에 의해 강력하고 지속적인 항종양 활성이 생성될 수 있음을 입증하며, 이는 주입된 제품의 양보다는 품질이 항종양 활성에 기여하는 데 더 중요하다는 것을 나타낸다.
인터루킨 (IL)-15는 림프구의 발생 및 지혈을 촉진시키는 사이토킨이다. IL-15의 증가된 수준은 T 세포 증식을 촉진시키고 T 세포 효과기 반응을 증진시킨다. 최근의 연구로부터의 자료는 IL-15가 항암 활성과 관련된 주요 인자들 중 하나인 기억 CD8 T 세포의 생성과 유지에 중요하다는 것을 보여주었다. IL-15는 T 세포 생존, 증식 및 기억 T 세포 생성과 같은 IL-15 매개 효과에 기여하는 IL-15 수용체 알파 사슬 (IL15RA 또는 RA라고도 함)에 결합한다.
IL-15RA는 T 세포 표면에서의 βγ 복합체에 결합하고 IL15는 T 세포 및 다른 유형의 세포의 표면에서의 이러한 IL-15RA/βγ 복합체와 결합함으로써 신호전달한다.
최근의 데이터는 IL-15 단독의 형질 감염이 T 세포 기능에 현저한 영향을 주지는 않지만 IL-15/IL-15RA의 형질 감염이 T 세포가 생존하고 자가적으로 증식하는 것을 가능하게 함을 보여 주었다.
IL-15 단독 투여의 효능은 유리 IL-15RA의 이용 가능성과 그것의 짧은 반감기에 의해 제한될 수 있다. 가용성 IL-15/RA 복합체의 투여는 생체내에서의 II-15 반감기 및 생체 이용률을 크게 향상시켰다. 따라서 IL-15 단독이 아닌 이 복합체로의 마우스의 처리는 기억 CD8 T 세포 및 NK 세포의 강력한 증식과 유지를 일으킨다. 최근의 연구는 스시 도메인 (sushi)이라고 불리는 IL-15RA의 세포외 영역의 일부가 그의 IL15와의 결합에 필요하다는 것을 보여주었다 (WEI 외, J. Immunol., vol.167(1), p:277-282, 2001). 링커를 함유하는 IL-15/RA 융합 단백질 또는 IL-15/스시(sushi) 융합 단백질은 IL-15 및 가용성 IL-15RA 단독보다 더 강력하다. IL-15/RA 또는 IL-15/스시(sushi)의 조합은 IL-15 활성을 최대화할 수 있다. 그러나, 삽입 서열 길이가 형질 감염 효율 및 유전자 발현 수준에 영향을 미칠 수 있기 때문에, CAR 및 IL-15/RA 또는 IL-15/스시(sushi) 둘 다를 같은 구축물에 통합시킨 디자인이 T 또는 NK 세포에서 그의 목적하는 생물학적 특성을 유지하는지에 대해서는 확실하지 않다.
본 개시내용은 단일 구축물 내에 CAR 및 IL-15/RA 또는 IL-15/스시 (sushi) 둘 모두를 갖는 조작된 세포를 제공한다. 일부 실시 양태에서, 본 개시내용은 더욱 높은 바이러스 역가를 생성하고 더욱 강력한 프로모터를 사용하여 CAR 및 IL-15/RA 또는 IL-15/스시(sushi) 둘 모두를 유도하는 방법들을 포함한다.
일부 실시 양태에서, 본 개시내용은 (1) CD4, CD2, CD3, CD7, CD5, CD45, CD20, CD19, CD33, CD123, CS1, 및 B-세포 성숙 항원 (BCMA)을 포함하나 이에 한정되지는 않는 항원을 표적으로 하는 CAR; 및 (2) IL-15; (3) IL-15RA (RA) 또는 스시 (sushi)를 갖는 조작된 세포를 제공한다. 추가의 실시 양태에서, CAR은 키메라 항원 수용체, CD28, CD2, 4-1BB 및 OX40과 같은 보조 자극 엔도 도메인 중 하나 이상 및 CD3 제타 사슬의 세포내 도메인을 포함한다. 추가의 실시 양태에서, 강력한 프로모터는 SFFV 일 수 있으나, 이에 한정되는 것은 아니다. CAR, IL-15/RA 또는 스시 및 유도성 자살 유전자 ("안전 스위치"), 또는 조합물은 렌티바이러스 벡터, 아데노바이러스 벡터 및 레트로바이러스 벡터와 같은 벡터 또는 플라스미드 상에 조립될 수 있다. "안전 스위치"의 도입은 안전 프로파일을 현저하게 증가시킬 수 있으며 CAR의 온-표적 또는 오프-종양 독성을 제한할 수 있다.
CD4IL15RA
-CAR의
특징화
CD4IL15RA-CAR을 생성하였으며 이는 IL15RA에 연결된 제3세대 CD4CAR을 포함한다 (도 34). CAR, (3세대), 스시/IL-15의 조합물은 발현 벡터 상에서 조립되며, 이들의 발현은 SFFV 프로모터에 의해 유도된다 (도 34). 스시/IL-15와 함께 CAR은 P2A 절단 서열과 연결되어 있다. 스시/IL-15 부분은 스시 도메인에 융합된 IL-2 신호 펩타이드로 구성되어 있으며 26-아미노산 폴리-프롤린 링커를 통해 IL-5에 연결된다 (도 34).
CD4IL15RA 구축물을 검증하기 위해, HEK293FT 세포를 GFP (대조) 또는 CD4IL15RA를 위한 렌티바이러스 플라스미드로 형질 감염시켰다. 형질 감염으로부터 약 60시간 후에, HEK-293FT 세포와 상청액 둘 모두를 수집하였다. 세포를 단백질분해효소 억제제 칵테일을 함유하는 RIPA 완충액에서 용해시키고 전기 영동하였다. 겔을 이모빌론(Immobilon) FL 블로팅 막에 옮기고, 차단하고, 마우스 항-인간 CD3z 항체로 1:500으로 프로빙하였다. 세척 후, 막을 염소 항-마우스 HRP 접합체로 프로빙하고, 세척하고, HyGlo HRP 기질로 처리한 후 필름에 노출시켰다. CD4IL15RA-CAR은 레인 3 (화살표)에서의 재조합 IL-15 단백질 옆에 도시된 바와 같이, HEK 293 세포 (레인 2, 도 35a)에서 성공적으로 발현되었다. 신선한 HEK-293 세포의 형질 도입으로CD4IL15RA-CAR 렌티바이러스 상청액을 추가로 검사하였다 (도 35a). GFP 또는 형질 감염된 HEK-293FT 세포로부터의 CD4IL15RA-CAR 바이러스 상청액으로 HEK-293 세포를 형질 도입시켰다. 폴리브렌을 4 uL/mL까지 첨가하였다. 16시간 후에 배지를 바꾸고 바이러스 상청액 또는 폴리브렌을 함유하지 않은 배지로 교체하였다. 형질 도입 3일 후에, 세포를 채취하고 염소-항-마우스 F(Ab')2 항체로 1:250에서 30분 동안 염색하였다. 세포를 세척하고 스트렙타비딘-PE (streptavidin-PE) 접합체로 1:500에서 염색하고, 세척하고, 2% 포르말린에 현탁시키고, 유동 세포측정법으로 분석하였다. 도 35b는 CD4IL15RA-CAR 렌티바이러스로 형질 도입된 HEK-293 세포가 F(Ab)2-PE에 대해 80% 양성이었지만 (동그라미로 표시됨, 도 35b) GFP 대조 렌티바이러스로의 형질 도입은 F(Ab)2-PE에 대해 최소였다 (도 35b, 왼쪽).
CD4IL15RA-CAR NK 세포의 생산
NK-92 세포를 CD4IL15RA-CAR 렌티바이러스 상청액으로 형질 도입시켰다. 5일간 인큐베이션 후에, 세포를 채취하고 염소 항-마우스 F(Ab')2와 1:250에서 30분 동안 인큐베이션하였다. 세포를 세척하고, 현탁시키고, 스트렙타비딘-PE (streptavidin-PE )로 30분 동안 염색하였다. 세포를 세척하고 2% 포르말린에 현탁시키고, 유동 세포측정법으로 분석하여, 형질 도입된 세포의 거의 70%가 CD4IL15RA-CAR을 발현하는 결과를 얻었다 (동그라미로 표시됨, 도 36). CD4IL15RA-CAR에 대한 추가 실험 검사는 시험관 내 및 생체내에서의 백혈병/림프종 살상 분석, 및 CD4CAR로 형질 도입된 세포와의 표적 살상 및 증식 속도의 비교를 포함할 것이다. 본 발명자는 또한 CD19IL15RA-CAR을 생성하기 위해 위에서 설명한 같은 전략을 사용하였다.
CD4IL15RA
-CAR T 세포의 생산
인간 제대 백혈구연층 세포를 CD4IL15RA-CAR 렌티바이러스 상청액으로 형질 도입시켰다. 5일간 인큐베이션 후에, 세포를 채취하고 염소 항-마우스 F(Ab')2와 1:250에서 30분 동안 인큐베이션하였다. 세포를 세척하고, 현탁시키고, 스트렙타비딘-PE (streptavidin-PE )로 30분 동안 염색하였다. 세포를 세척하고 2% 포르말린에 현탁시키고, 유동 세포측정법으로 분석하여, 형질 도입된 세포의 63%가 CD4IL15RA-CAR을 발현하는 결과를 얻었다 (동그라미로 표시됨, 도 37). CD4IL15RA-CAR에 대한 추가 실험 검사는 시험관 내 및 생체내에서의 백혈병/림프종 살상 분석, 및 CD4CAR로 형질 도입된 세포와의 표적 살상 및 증식 속도의 비교를 포함할 것이다.
CD4IL15RACAR
NK
세포를 이하 CD4 양성 세포주:
Karpas
299 및
MOLT4와
공동 배양하는 것에 의해 시험관 내에서
CD4CAR
NK
세포와 비교하여 항-백혈병 활성을 검사하였다.
Karpas 299 세포주는 역형성 거대 T 세포 림프종을 가진 환자로부터 유래되었다. CD4를 발현하는 MOLT4 세포주는 급성 림프모구성 백혈병 (T-ALL)을 가진 19세 환자의 말초 혈액으로부터 확립되었다. 4시간의 공동 배양 실험동안, CD4IL15RA CAR NK 세포는 효과기:표적의 5:1 비율에서, Karpas 299 세포를 CD4CAR NK 세포의 것 (82%, 도 38)보다 더 높은 비율로 엄청난 살상을 나타냈다 (95%). 유사하게, MOLT4 세포와 1:1로 공동 배양하였을 때, CD4IL15RA CAR NK 세포는 밤샘 분석에서 CD4CAR NK 세포보다 높은 비율 (84% 대 65%)로 표적 세포를 용해시켰다 (도 39). 이런 결과는 CD4IL15 CAR NK 세포가 적어도 CD4CAR NK 세포뿐만 아니라 적어도 종양 세포를 제거할 수 있음을 보여준다.
CD4CAR
및
CD4IL15RA
CAR T 세포는
CD4CAR보다
생체내에서
더 강력한 항-종양 활성을
나타낸다
CD4CAR 및 CD4IL15RACAR T 세포의 생체내 항종양 활성을 평가하고, CD4CAR T 세포와 비교하여 CD4IL15RA CAR T 세포 지속성에 있어서 가능한 증가를 결정하기 위해, NSG 마우스를 사용하여 측정 가능한 종양 형성을 유도하기 위해, 치사량에 가까운 방사선 조사하고 100% CD4인 급성 골수성 백혈병 세포주 (M5)인 루시페레이스-발현 MOLM13 세포를 정맥내 주사하여 이종 마우스 모델을 개발하였다. 종양 세포 주사 3일 후, 6마리의 마우스 각각에게 8 x 106 CD4CAR, CD4IL15RACAR T 세포 또는 벡터 대조 T 세포를 정맥내 주사하였다. 제3, 6, 9 및 11일째에, 마우스에게 RediJect D-Luciferin (Perkin Elmer)을 피하 주사하고 IVIS 영상을 시행하여 종양 부하를 측정했다 (도 40). CD4CAR T 세포-처리된 마우스는 6일째에 대조에 비해 52%의 낮은 종양 부담을 가진 반면, CD4IL15RA CAR T 세포-처리된 마우스는 74%의 낮은 종양 부담을 가졌다 (도 41). 11일째, 거의 모든 종양 세포가 이 두 그룹 모두에서 용해되었다. 독립표본 T 검정 분석은 대조와 비교시 CD4CAR 및 CD4IL15RACAR T 세포 처리 그룹에서 9일까지 더 적은 광 강도, 따라서 더 적은 종양 부하를 가지는 것으로 두 그룹 사이에 매우 유의한 차이 (P=0.0045)를 드러냈다.
GFP
리포터를 이용한 프로모터 테스트
HEK293FT 세포를 SFFV, EF1 또는 CAG 프로모터 하에서 GFP를 발현하는 렌티바이러스 플라스미드로 형질 감염시켰다. 형질 감염 대략 60시간 후에 각각으로부터 상청액을 수집하였다. 상대적인 바이러스 역가를 먼저 3개 프로모터 각자로부터의 상청액으로 HEK293 세포를 형질 도입하는 것에 의해 측정하였다. HEK-293 세포를 3개 형질 감염된 HEK-293FT 세포 각자로부터의 GFP 바이러스 상청액으로 형질 도입시켰다. 폴리브렌을 4 uL/mL까지 첨가하였다. 16시간 후에 배지를 바꾸고 바이러스 상청액 또는 폴리브렌을 함유하고 있지 않은 배지로 교체하였다. 형질 도입 3일 후, 세포를 채취하고 세척하고, 2% 포르말린에 현탁시키고, GFP 발현 (FITC)에 대해 유동 세포측정법으로 분석하였다. GFP 발현은 각 샘플에서 나타났지만, SFFV 프로모터를 사용하여 만든 바이러스로 형질 도입된 세포에서 가장 높았다.
활성화된 인간 제대 백혈구연층 세포를 각 프로모터로부터의 GFP 렌티바이러스 상청액 (HEK293 형질 도입 효율의 결과에 기초한 양)으로 형질 도입시켰다. 인큐베이션 5일 후, 세포를 채취하고, 세척하고 2% 포르말린에서 현탁시키고, GFP 발현에 대해 유동 세포측정법으로 분석하였다. 43%의 세포가 높은 수준 (>103)으로 GFP를 발현한 반면, 프로모터 EF1를 사용하여 바이러스로 형질 도입된 세포에 대한 GFP 발현 (15%) 및 CAG를 사용하여 바이러스로 형질 도입된 세포에 대한 GFP 발현 (3%)은 상당히 낮았다. 5일 후, 같은 방법으로 분석된 세포는 각각에 대해 거의 동일한 백분율을 나타냈다 (각기 46%, 15% 및 3%; 도 43). 이런 결과는 SFFV 프로모터가 EF1 또는 CAG 프로모터보다 더 강한 발현을 일으키고, 발현이 형질 도입 후 적어도 10일 동안 높게 유지된다는 것을 나타낸다. 추가적인 실험 테스트는 형질 도입된 세포에 대한 10일 윈도우 이상으로 더 긴 인큐베이션 시간을 포함할 것이다.
T 항원 인식 모이어티 (CD4, CD8, CD3, CD5, CD7, 및 CD2 중 적어도 하나, 또는 이들의 일부 또는 이들의 조합), 힌지 영역 및 T 세포 활성화 도메인 중 적어도 하나를 포함하는 CAR 유전자를 생성하는 방법들이 제공된다.
CD33, CD123, CD19, CD20, CD22, CD269, CS1, CD38, CD52, ROR1, PSMA, CD138, 및 GPC3 중 적어도 하나 또는 하나의 힌지 영역 및 T 세포 활성화 도메인들의 일부 또는 조합을 포함하는 항원(들)을 표적으로 하는 CAR (cCAR)의 다중 단위를 생성하는 방법들이 제공된다. 본원에 인용 및/또는 개시된 모든 참고 문헌은 그 전문이 본원에 참조로 포함된다.
제공된 방법은 또한 1) 백혈병 및 림프종을 발현하는 CD45를 표적화하고 자가 살상을 회피하는 CAR T 또는 NK 세포의 생성; 2) 억제 종양 미세환경을 극복하고 증진된 항종양 활성 및 장기적 지속을 나타내도록 설계된 "갑옷을 입은" CAR T 또는 NK 세포의 생성을 포함한다. 본 발명은 위에서 설명되고 예시된 실시 양태들로 제한되지는 않으나, 첨부된 청구항들의 범위 내에서 변형 및 수정을 할 수 있다. 특허, 공개된 출원, 기술적 논문 및 학술 자료를 비롯한 다양한 공개가 본 명세서 전반에 걸쳐 인용된다. 각각의 인용된 공개는 모든 목적을 위해 그 전체가 여기에 참고로 포함된다. 본 발명의 양태와 관련되는 다양한 용어는 본 명세서 및 청구항의 전반에 걸쳐 사용된다. 이러한 용어는 달리 나타내지 않는 한 당해 기술 분야에서의 통상적인 그들의 의미가 제공되어야 한다. 다른 특별히 정의된 용어는 본원에 제공된 정의와 일치하는 방식으로 해석되어야 한다.
상청액에서의 바이러스 벡터 입자의 기능적 역가 (유동 세포측정법으로 측정된 바와 같은 % GFP 세포는 더 높은 역가 바이러스가 더 많은 세포에 침투하여 더 높은 % GFP 세포 집단을 초래하기 때문에 프록시 바이러스 역가 조정을 가능케 한다).
상청액 각각에서의 바이러스 벡터 입자의 기능적인 역가를 측정하기 위해, HEK 293 세포를 12 웰(well) 조직-배양 처리된 플레이트의 웰 (well)당 30μL (저용량), 125μL (중간용량), 또는 500μL (고용량)의 EF1-GFP 또는 SFFV-GFP 바이러스 상청액으로 형질 도입시켰다. 다음날 아침, 배양 배지를 DMEM+10% FBS로 바꾸었다.
이어서, 형질 도입된 세포를 트립신처리하고, 세척하고, 포르말린에 현탁시키고, 유동 세포측정법 분석에 맡겼다. 각 조건에서의 GFP+ 세포의 백분율을 FITC 채널을 사용하여 유동 세포측정법에 의해 측정하였다 (도 43). 각각의 경우에, GFP+의 백분율은 상응하는 용량의 EF1-GFP 바이러스 상청액으로 형질 도입된 세포보다 SFFV-GFP로 형질 도입된 세포에서 더 높았다(저용량의 경우 50% 대 18%, 중간용량의 경우 80% 대 40%, 및 고용량의 경우 82% 대 70%). 이것으로부터, 역가의 측면에서, 최고 분량의 EF1-프로모터 바이러스를 사용하는 것이 최저 분량의 SFFV- 프로모터 바이러스를 사용하는 것에 필적하고, 후속 형질 도입 실험에 대한 상대적 프로모터 강도의 비교를 가능케 할 것임을 알아냈다.
형질 도입된 세포를 또한 각 웰 (well) 에 대해 같은 노출 조건에서 GFP를 사용하여 20x에서 EVOS 형광 현미경으로 시각화시켰다 (도 42). SFFV-GFP 바이러스 상청액으로 형질 도입된 세포는 EF1-GFP로 형질 도입된 세포보다 획기적으로 더 밝았다. 또한, 높은 바이러스 용량 부하 하에서의 EF1-프로모터 이미지와 낮은 바이러스 용량 부하를 사용한 SFFV-프로모터 이미지의 비교는 유사한 형광 강도를 보여준다. 이것은 SFFV 프로모터가 유전자 발현의 더 강력한 유도체임을 시사한다.
1차 T 세포에서의 표면 발현 및 상이한 프로모터의 지속성 비교
(
T 세포 형질
도입에 대한 유동 세포측정법으로
측정된
%GFP
세포는
HEK293
세포에 관한 이전 실험으로부터 기대되는 바와 같이 GFP 세포 집단에서의 기대되는 차이를 나타낸다)
1차 T 세포에서 프로모터 형질 도입 효율 및 표면 발현의 지속성을 측정하기 위해, 레트로넥틴(Clontech)으로 미리 코팅된 12-웰 (well) 조직 배양 처리된 플레이트에서, 활성화된 제대혈 백혈구연층 T 세포를 50μL SFFV-GFP 또는 1 mL EF1-GFP 바이러스 상청액으로 형질 도입시켰다. 두 번의 밤샘 형질 도입 후, 세포를 300 IU/mL IL-2 (Peprotech)를 함유하는 T 세포 배지에서 배양하고 1.0-4.0 x 106/mL로 유지했다. 세포를 세척하고, 포르말린에 현탁시키고, 형질 도입 후 7, 14, 21 및 28일에 FITC 채널을 사용하여 유동 세포측정법 분석을 수행하여 GFP+ 세포의 백분율을 측정하였다. 전체 GFP+ 세포의 백분율이 이 기간에 걸쳐 감소하더라도, GFP+ 세포의 백분율은 EF1-GFP-형질 도입된 T 세포와 비교하여 SFFV-GFP로 형질 도입된 T 세포에 대해 지속해서 더 높았다 (도 44A). 추가 비교는 더 많은 양 (1mL)의 EF1-GFP 상청액으로 형질 도입된 T 세포의 백분율은 사실상 더 적은 양 (50μL, 또는 20배 적은)의 SFFV-GFP로 형질 도입된 GFP+ 세포의 백분율에 비해 제7일 내지 제28일에 60% 이상에서 40% 이하로 감소하였음을 나타냈다 (도 44B). 이것은 SFFV 프로모터를 이용한 형질 도입이 형질 도입된 세포의 더 오랜 지속성을 초래한다는 것을 시사한다.
BCMA
또는
TACI
또는
BAFF
-R CAR 항원 중 적어도 하나를 발현하는 세포를 표적으로 하는
BCMA
또는
TACI
또는
BAFF
-R CAR
NK
세포 혹은 T 세포
BCMA 또는 TACI 또는 BAFF-R 중 적어도 하나를 표적으로 하는 CAR NK 세포 혹은 T 세포의 세포독성 능력을 평가하기 위해, BCMA 또는 TACI 또는 BAFF-R 중 적어도 하나를 발현하는 세포주 또는 1차 인간 세포로 공동 배양 분석을 수행한다. 표적 세포를 용해시키는 상기 언급된 CAR NK 세포 또는 T 세포의 능력을 유동 세포측정법 분석으로 정량화하고 표적 세포를 사이토트래커 염료 (CMTMR)로 염색하였다. 24 시간의 긴 배양에서 용해를 관찰한다.
BCMA
또는
TACI
또는
BAFF
-R 항원 중 적어도 하나를 발현하는 세포를 표적으로 하는
BAFF
또는
APRIL
CAR
NK
또는 T 세포.
CAR에서의 키메라 항원 수용체는 BCMA 또는 TACI 또는 BAFF-R의 리간드이다.
BCMA 또는 TACI 또는 BAFF-R 중 적어도 하나를 표적으로 하는 CAR NK 또는 T 세포의 세포독성 능력을 평가하기 위해, BCMA 또는 TACI 또는 BAFF-R 중 적어도 하나를 발현하는 세포주 또는 1차 인간 세포로 공동 배양 분석을 수행한다. 표적 세포를 용해시키는 상기 언급된 CAR NK 또는 T 세포의 능력을 유동 세포측정법 분석으로 정량화하고 표적 세포를 사이토트래커 염료 (CMTMR)로 염색하였다. 24 시간의 긴 배양에서 용해를 관찰한다.
참고문헌
서열 목록의 포함
본 출원을 위한 서열 목록(Sequence Listing)은 그 전체가 본원에 참조로 포함된다. 서열 목록(Sequence Listing)은 2016년 6월 24일에 작성된 "sequence_listing.txt"라는 제목의 컴퓨터 판독 가능한 ASCII 텍스트 파일에 개시된다. sequence-listing.txt 파일은 크기가 140KB이다.
SEQUENCE LISTING
<110> iCell Gene Therapeutics LLC
<120> Chimeric Antigen Receptors (CARs), Compositions and
Methods of Use
<130> 2541-3 PCT
<140> 0000000
<141> 2016-06-24
<150> 62/184,321
<151> 2015-06-25
<160> 36
<170> PatentIn version 3.5
<210> 1
<211> 830
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 1
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu
20 25 30
Ala Val Ser Leu Gly Glu Arg Val Thr Met Asn Cys Lys Ser Ser Gln
35 40 45
Ser Leu Leu Tyr Ser Thr Asn Gln Lys Asn Tyr Leu Ala Trp Tyr Gln
50 55 60
Gln Lys Pro Gly Gln Ser Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr
65 70 75 80
Arg Glu Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr
85 90 95
Asp Phe Thr Leu Thr Ile Ser Ser Val Gln Ala Glu Asp Val Ala Val
100 105 110
Tyr Tyr Cys Gln Gln Tyr Tyr Ser Tyr Arg Thr Phe Gly Gly Gly Thr
115 120 125
Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
130 135 140
Gly Gly Gly Ser Gln Val Gln Leu Gln Gln Ser Gly Pro Glu Val Val
145 150 155 160
Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr
165 170 175
Phe Thr Ser Tyr Val Ile His Trp Val Arg Gln Lys Pro Gly Gln Gly
180 185 190
Leu Asp Trp Ile Gly Tyr Ile Asn Pro Tyr Asn Asp Gly Thr Asp Tyr
195 200 205
Asp Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Thr Ser Thr
210 215 220
Ser Thr Ala Tyr Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala
225 230 235 240
Val Tyr Tyr Cys Ala Arg Glu Lys Asp Asn Tyr Ala Thr Gly Ala Trp
245 250 255
Phe Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Thr Thr
260 265 270
Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln
275 280 285
Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala
290 295 300
Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala
305 310 315 320
Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr
325 330 335
Leu Tyr Cys Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met
340 345 350
Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro
355 360 365
Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Lys Arg Gly Arg
370 375 380
Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln
385 390 395 400
Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu
405 410 415
Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala
420 425 430
Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu
435 440 445
Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp
450 455 460
Pro Glu Met Gly Gly Lys Pro Gln Arg Arg Lys Asn Pro Gln Glu Gly
465 470 475 480
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
485 490 495
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
500 505 510
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
515 520 525
Met Gln Ala Leu Pro Pro Arg Gly Ser Gly Ala Thr Asn Phe Ser Leu
530 535 540
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Tyr Arg
545 550 555 560
Met Gln Leu Leu Ser Cys Ile Ala Leu Ser Leu Ala Leu Val Thr Asn
565 570 575
Ser Gly Ile His Val Phe Ile Leu Gly Cys Phe Ser Ala Gly Leu Pro
580 585 590
Lys Thr Glu Ala Asn Trp Val Asn Val Ile Ser Asp Leu Lys Lys Ile
595 600 605
Glu Asp Leu Ile Gln Ser Met His Ile Asp Ala Thr Leu Tyr Thr Glu
610 615 620
Ser Asp Val His Pro Ser Cys Lys Val Thr Ala Met Lys Cys Phe Leu
625 630 635 640
Leu Glu Leu Gln Val Ile Ser Leu Glu Ser Gly Asp Ala Ser Ile His
645 650 655
Asp Thr Val Glu Asn Leu Ile Ile Leu Ala Asn Asn Ser Leu Ser Ser
660 665 670
Asn Gly Asn Val Thr Glu Ser Gly Cys Lys Glu Cys Glu Glu Leu Glu
675 680 685
Glu Lys Asn Ile Lys Glu Phe Leu Gln Ser Phe Val His Ile Val Gln
690 695 700
Met Phe Ile Asn Thr Ser Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser
705 710 715 720
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Ser Leu Gln
725 730 735
Ala Pro Arg Arg Ala Arg Gly Cys Arg Thr Leu Gly Leu Pro Ala Leu
740 745 750
Leu Leu Leu Leu Leu Leu Arg Pro Pro Ala Thr Arg Gly Ile Thr Cys
755 760 765
Pro Pro Pro Met Ser Val Glu His Ala Asp Ile Trp Val Lys Ser Tyr
770 775 780
Ser Leu Tyr Ser Arg Glu Arg Tyr Ile Cys Asn Ser Gly Phe Lys Arg
785 790 795 800
Lys Ala Gly Thr Ser Ser Leu Thr Glu Cys Val Leu Asn Lys Ala Thr
805 810 815
Asn Val Ala His Trp Thr Thr Pro Ser Leu Lys Cys Ile Arg
820 825 830
<210> 2
<211> 2509
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 2
gcgatcgcat ggccttacca gtgaccgcct tgctcctgcc gctggccttg ctgctccacg 60
ccgccaggcc ggacatcgtg atgacccaaa gccccgacag cctggccgtg agcctgggcg 120
agagggtgac catgaactgc aaaagcagcc agtccctgct gtactccacc aaccagaaga 180
actacctggc ttggtatcaa cagaagcccg gacagagccc caagctgctg atctattggg 240
ccagcactag ggaaagcggc gtgcccgata ggttcagcgg cagcgggagc ggcacagact 300
tcactctgac cattagcagc gtgcaggctg aggatgtggc cgtctactac tgccagcagt 360
actacagcta caggaccttt gggggcggaa ctaagctgga gatcaaggga ggggggggat 420
ccgggggagg aggctccggc ggaggcggaa gccaagtgca actgcagcag agcggcccag 480
aggtggtcaa acctggggca agcgtgaaga tgagctgcaa ggctagcggc tataccttca 540
ccagctatgt gatccactgg gtgaggcaga aaccaggaca gggcctggac tggatcggct 600
acatcaaccc ctacaatgac ggcaccgatt atgacgaaaa attcaagggg aaggccaccc 660
tgaccagcga caccagcaca agcaccgcct acatggagct gtccagcctg aggtccgagg 720
acaccgccgt gtattactgt gccagggaga aggacaatta cgccaccggc gcttggttcg 780
cctactgggg ccagggcaca ctggtgacag tgagcagcac cacgacgcca gcgccgcgac 840
caccaacacc ggcgcccacc atcgcgtcgc agcccctgtc cctgcgccca gaggcgtgcc 900
ggccagcggc ggggggcgca gtgcacacga gggggctgga cttcgcctgt gatatctaca 960
tctgggcgcc cttggccggg acttgtgggg tccttctcct gtcactggtt atcacccttt 1020
actgcaggag taagaggagc aggctcctgc acagtgacta catgaacatg actccccgcc 1080
gccccgggcc cacccgcaag cattaccagc cctatgcccc accacgcgac ttcgcagcct 1140
atcgctccaa acggggcaga aagaaactcc tgtatatatt caaacaacca tttatgagac 1200
cagtacaaac tactcaagag gaagatggct gtagctgccg atttccagaa gaagaagaag 1260
gaggatgtga actgagagtg aagttcagca ggagcgcaga cgcccccgcg taccagcagg 1320
gccagaacca gctctataac gagctcaatc taggacgaag agaggagtac gatgttttgg 1380
acaagagacg tggccgggac cctgagatgg ggggaaagcc gcagagaagg aagaaccctc 1440
aggaaggcct gtacaatgaa ctgcagaaag ataagatggc ggaggcctac agtgagattg 1500
ggatgaaagg cgagcgccgg aggggcaagg ggcacgatgg cctttaccag ggtctcagta 1560
cagccaccaa ggacacctac gacgcccttc acatgcaggc cctgccccct cgcggaagcg 1620
gagccaccaa cttcagcctg ctgaagcagg ccggcgacgt ggaggagaac cccggcccca 1680
tgtacagaat gcagctgctg agctgcatcg ccctgagcct ggccctggtg accaacagcg 1740
gcatccacgt gttcatcctg ggctgcttca gcgccggcct gcccaagacc gaggccaact 1800
gggtgaacgt gatcagcgac ctgaagaaga tcgaggacct gatccagagc atgcacatcg 1860
acgccaccct gtacaccgag agcgacgtgc accccagctg caaggtgacc gccatgaagt 1920
gcttcctgct ggagctgcag gtgatcagcc tggagagcgg cgacgccagc atccacgaca 1980
ccgtggagaa cctgatcatc ctggccaaca acagcctgag cagcaacggc aacgtgaccg 2040
agagcggctg caaggagtgc gaggagctgg aggagaagaa catcaaggag ttcctgcaga 2100
gcttcgtgca catcgtgcag atgttcatca acaccagctc cggcggcggc tccggcggcg 2160
gcggctccgg cggcggcggc tccggcggcg gcggctccgg cggcggctcc ctgcaggccc 2220
ccagaagagc cagaggctgc agaaccctgg gcctgcccgc cctgctgctg ctgctgctgc 2280
tgagaccccc cgccaccaga ggcatcacct gccccccccc catgagcgtg gagcacgccg 2340
acatctgggt gaagagctac agcctgtaca gcagagagag atacatctgc aacagcggct 2400
tcaagagaaa ggccggcacc agcagcctga ccgagtgcgt gctgaacaag gccaccaacg 2460
tggcccactg gaccaccccc agcctgaagt gcatcagata agtttaaac 2509
<210> 3
<211> 995
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 3
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Asp Ile Gln Met Thr Gln Thr Thr Ser Ser Leu
20 25 30
Ser Ala Ser Leu Gly Asp Arg Val Thr Ile Ser Cys Arg Ala Ser Gln
35 40 45
Asp Ile Ser Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Asp Gly Thr
50 55 60
Val Lys Leu Leu Ile Tyr His Thr Ser Arg Leu His Ser Gly Val Pro
65 70 75 80
Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Ser Leu Thr Ile
85 90 95
Ser Asn Leu Glu Gln Glu Asp Ile Ala Thr Tyr Phe Cys Gln Gln Gly
100 105 110
Asn Thr Leu Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Thr
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
130 135 140
Val Lys Leu Gln Glu Ser Gly Pro Gly Leu Val Ala Pro Ser Gln Ser
145 150 155 160
Leu Ser Val Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly
165 170 175
Val Ser Trp Ile Arg Gln Pro Pro Arg Lys Gly Leu Glu Trp Leu Gly
180 185 190
Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Asn Ser Ala Leu Lys Ser
195 200 205
Arg Leu Thr Ile Ile Lys Asp Asn Ser Lys Ser Gln Val Phe Leu Lys
210 215 220
Met Asn Ser Leu Gln Thr Asp Asp Thr Ala Ile Tyr Tyr Cys Ala Lys
225 230 235 240
His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly
245 250 255
Thr Ser Val Thr Val Ser Ser Thr Thr Thr Pro Ala Pro Arg Pro Pro
260 265 270
Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu
275 280 285
Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp
290 295 300
Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly
305 310 315 320
Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg
325 330 335
Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln
340 345 350
Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu
355 360 365
Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala
370 375 380
Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu
385 390 395 400
Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp
405 410 415
Pro Glu Met Gly Gly Lys Pro Gln Arg Arg Lys Asn Pro Gln Glu Gly
420 425 430
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
435 440 445
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
450 455 460
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
465 470 475 480
Met Gln Ala Leu Pro Pro Arg Gly Ser Gly Ala Thr Asn Phe Ser Leu
485 490 495
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ala Leu
500 505 510
Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu His Ala Ala
515 520 525
Arg Pro Gln Ile Val Leu Ser Gln Ser Pro Ala Ile Leu Ser Ala Ser
530 535 540
Pro Gly Glu Lys Val Thr Met Thr Cys Arg Ala Ser Ser Ser Val Ser
545 550 555 560
Tyr Ile His Trp Phe Gln Gln Lys Pro Gly Ser Ser Pro Lys Pro Trp
565 570 575
Ile Tyr Ala Thr Ser Asn Leu Ala Ser Gly Val Pro Val Arg Phe Ser
580 585 590
Gly Ser Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Arg Val Glu
595 600 605
Ala Glu Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Trp Thr Ser Asn Pro
610 615 620
Pro Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly
625 630 635 640
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Gln
645 650 655
Gln Pro Gly Ala Glu Leu Val Lys Pro Gly Ala Ser Val Lys Met Ser
660 665 670
Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr Asn Met His Trp Val
675 680 685
Lys Gln Thr Pro Gly Arg Gly Leu Glu Trp Ile Gly Ala Ile Tyr Pro
690 695 700
Gly Asn Gly Asp Thr Ser Tyr Asn Gln Lys Phe Lys Gly Lys Ala Thr
705 710 715 720
Leu Thr Ala Asp Lys Ser Ser Ser Thr Ala Tyr Met Gln Leu Ser Ser
725 730 735
Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys Ala Arg Ser Thr Tyr
740 745 750
Tyr Gly Gly Asp Trp Tyr Phe Asn Val Trp Gly Ala Gly Thr Thr Val
755 760 765
Thr Val Ser Ala Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala
770 775 780
Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg
785 790 795 800
Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys
805 810 815
Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu
820 825 830
Leu Ser Leu Val Ile Thr Leu Tyr Cys Arg Ser Lys Arg Ser Arg Leu
835 840 845
Leu His Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr
850 855 860
Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr
865 870 875 880
Arg Ser Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln
885 890 895
Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu
900 905 910
Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly
915 920 925
Gly Lys Pro Gln Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu
930 935 940
Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys
945 950 955 960
Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu
965 970 975
Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu
980 985 990
Pro Pro Arg
995
<210> 4
<211> 3004
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 4
gcgatcgcat ggccttacca gtgaccgcct tgctcctgcc gctggccttg ctgctccacg 60
ccgccaggcc ggacatccag atgacacaga ctacatcctc cctgtctgcc tctctgggag 120
acagagtcac catcagttgc agggcaagtc aggacattag taaatattta aattggtatc 180
agcagaaacc agatggaact gttaaactcc tgatctacca tacatcaaga ttacactcag 240
gagtcccatc aaggttcagt ggcagtgggt ctggaacaga ttattctctc accattagca 300
acctggagca agaagatatt gccacttact tttgccaaca gggtaatacg cttccgtaca 360
cgttcggagg ggggaccaag ctggagatca caggtggcgg tggctcgggc ggtggtgggt 420
cgggtggcgg cggatctgag gtgaaactgc aggagtcagg acctggcctg gtggcgccct 480
cacagagcct gtccgtcaca tgcactgtct caggggtctc attacccgac tatggtgtaa 540
gctggattcg ccagcctcca cgaaagggtc tggagtggct gggagtaata tggggtagtg 600
aaaccacata ctataattca gctctcaaat ccagactgac catcatcaag gacaactcca 660
agagccaagt tttcttaaaa atgaacagtc tgcaaactga tgacacagcc atttactact 720
gtgccaaaca ttattactac ggtggtagct atgctatgga ctactggggc caaggaacct 780
cagtcaccgt ctcctcaacc acgacgccag cgccgcgacc accaacaccg gcgcccacca 840
tcgcgtcgca gcccctgtcc ctgcgcccag aggcgtgccg gccagcggcg gggggcgcag 900
tgcacacgag ggggctggac ttcgcctgtg atatctacat ctgggcgccc ttggccggga 960
cttgtggggt ccttctcctg tcactggtta tcacccttta ctgcaaacgg ggcagaaaga 1020
aactcctgta tatattcaaa caaccattta tgagaccagt acaaactact caagaggaag 1080
atggctgtag ctgccgattt ccagaagaag aagaaggagg atgtgaactg agagtgaagt 1140
tcagcaggag cgcagacgcc cccgcgtacc agcagggcca gaaccagctc tataacgagc 1200
tcaatctagg acgaagagag gagtacgatg ttttggacaa gagacgtggc cgggaccctg 1260
agatgggggg aaagccgcag agaaggaaga accctcagga aggcctgtac aatgaactgc 1320
agaaagataa gatggcggag gcctacagtg agattgggat gaaaggcgag cgccggaggg 1380
gcaaggggca cgatggcctt taccagggtc tcagtacagc caccaaggac acctacgacg 1440
cccttcacat gcaggccctg ccccctcgcg gaagcggagc caccaacttc agcctgctga 1500
agcaggccgg cgacgtggag gagaaccccg gccccatggc cttaccagtg accgccttgc 1560
tcctgccgct ggccttgctg ctccacgccg ccaggccgca gatcgtgctg agccagagcc 1620
ctgccatcct gtccgcaagc ccaggcgaga aggtgaccat gacctgtagg gccagcagct 1680
ccgtgagcta catccactgg tttcagcaga agcctggaag cagccctaag ccctggatct 1740
acgccacaag caatctggct agcggcgtgc ccgtgaggtt cagcggcagc gggagcggga 1800
ccagctacag cctgactatc agcagggtgg aggccgagga cgccgccaca tactactgcc 1860
aacagtggac ctccaaccca cccacctttg gaggagggac aaaactggag atcaaagggg 1920
gcggagggtc cggaggcggc ggaagcgggg gagggggaag ccaggtccaa ctgcaacagc 1980
ccggagcaga actggtcaaa ccaggcgcca gcgtgaagat gagctgcaag gccagcgggt 2040
acaccttcac ttcctataac atgcactggg tgaagcagac cccaggaagg ggcctggagt 2100
ggatcggggc aatctatccc ggcaacggcg acacaagcta caaccagaag ttcaagggga 2160
aagccactct gaccgccgac aagtccagct ccaccgccta catgcagctg agctccctga 2220
ccagcgagga cagcgccgtg tactattgcg ccagaagcac ttattacgga ggggactggt 2280
acttcaacgt gtggggggca gggaccaccg tgaccgtgtc cgccaccacg acgccagcgc 2340
cgcgaccacc aacaccggcg cccaccatcg cgtcgcagcc cctgtccctg cgcccagagg 2400
cgtgccggcc agcggcgggg ggcgcagtgc acacgagggg gctggacttc gcctgtgata 2460
tctacatctg ggcgcccttg gccgggactt gtggggtcct tctcctgtca ctggttatca 2520
ccctttactg caggagtaag aggagcaggc tcctgcacag tgactacatg aacatgactc 2580
cccgccgccc cgggcccacc cgcaagcatt accagcccta tgccccacca cgcgacttcg 2640
cagcctatcg ctccagagtg aagttcagca ggagcgcaga cgcccccgcg taccagcagg 2700
gccagaacca gctctataac gagctcaatc taggacgaag agaggagtac gatgttttgg 2760
acaagagacg tggccgggac cctgagatgg ggggaaagcc gcagagaagg aagaaccctc 2820
aggaaggcct gtacaatgaa ctgcagaaag ataagatggc ggaggcctac agtgagattg 2880
ggatgaaagg cgagcgccgg aggggcaagg ggcacgatgg cctttaccag ggtctcagta 2940
cagccaccaa ggacacctac gacgcccttc acatgcaggc cctgccccct cgctaagttt 3000
aaac 3004
<210> 5
<211> 1001
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 5
Asp Arg Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu
1 5 10 15
Leu Leu His Ala Ala Arg Pro Asp Ile Gln Met Thr Gln Thr Thr Ser
20 25 30
Ser Leu Ser Ala Ser Leu Gly Asp Arg Val Thr Ile Ser Cys Arg Ala
35 40 45
Ser Gln Asp Ile Ser Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Asp
50 55 60
Gly Thr Val Lys Leu Leu Ile Tyr His Thr Ser Arg Leu His Ser Gly
65 70 75 80
Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Ser Leu
85 90 95
Thr Ile Ser Asn Leu Glu Gln Glu Asp Ile Ala Thr Tyr Phe Cys Gln
100 105 110
Gln Gly Asn Thr Leu Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu
115 120 125
Ile Thr Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
130 135 140
Ser Glu Val Lys Leu Gln Glu Ser Gly Pro Gly Leu Val Ala Pro Ser
145 150 155 160
Gln Ser Leu Ser Val Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp
165 170 175
Tyr Gly Val Ser Trp Ile Arg Gln Pro Pro Arg Lys Gly Leu Glu Trp
180 185 190
Leu Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Asn Ser Ala Leu
195 200 205
Lys Ser Arg Leu Thr Ile Ile Lys Asp Asn Ser Lys Ser Gln Val Phe
210 215 220
Leu Lys Met Asn Ser Leu Gln Thr Asp Asp Thr Ala Ile Tyr Tyr Cys
225 230 235 240
Ala Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly
245 250 255
Gln Gly Thr Ser Val Thr Val Ser Ser Thr Thr Thr Pro Ala Pro Arg
260 265 270
Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg
275 280 285
Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly
290 295 300
Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr
305 310 315 320
Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg
325 330 335
Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro
340 345 350
Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu
355 360 365
Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala
370 375 380
Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu
385 390 395 400
Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly
405 410 415
Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu
420 425 430
Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser
435 440 445
Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly
450 455 460
Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu
465 470 475 480
His Met Gln Ala Leu Pro Pro Arg Gly Ser Gly Ala Thr Asn Phe Ser
485 490 495
Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ala
500 505 510
Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu His Ala
515 520 525
Ala Arg Pro Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala
530 535 540
Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ser Ser Gln Ser Ile
545 550 555 560
Val His Ser Val Gly Asn Thr Phe Leu Glu Trp Tyr Gln Gln Lys Pro
565 570 575
Gly Lys Ala Pro Lys Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser
580 585 590
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
595 600 605
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
610 615 620
Phe Gln Gly Ser Gln Phe Pro Tyr Thr Phe Gly Gln Gly Thr Lys Val
625 630 635 640
Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
645 650 655
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro
660 665 670
Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Tyr Glu Phe Ser
675 680 685
Arg Ser Trp Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
690 695 700
Trp Val Gly Arg Ile Tyr Pro Gly Asp Gly Asp Thr Asn Tyr Ser Gly
705 710 715 720
Lys Phe Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn Thr
725 730 735
Ala Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
740 745 750
Tyr Cys Ala Arg Asp Gly Ser Ser Trp Asp Trp Tyr Phe Asp Val Trp
755 760 765
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Thr Thr Thr Pro Ala Pro
770 775 780
Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu
785 790 795 800
Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg
805 810 815
Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly
820 825 830
Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys
835 840 845
Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg
850 855 860
Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro
865 870 875 880
Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser
885 890 895
Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu
900 905 910
Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg
915 920 925
Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln
930 935 940
Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr
945 950 955 960
Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp
965 970 975
Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala
980 985 990
Leu His Met Gln Ala Leu Pro Pro Arg
995 1000
<210> 6
<211> 3016
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 6
gcgatcgcat ggccttacca gtgaccgcct tgctcctgcc gctggccttg ctgctccacg 60
ccgccaggcc ggacatccag atgacacaga ctacatcctc cctgtctgcc tctctgggag 120
acagagtcac catcagttgc agggcaagtc aggacattag taaatattta aattggtatc 180
agcagaaacc agatggaact gttaaactcc tgatctacca tacatcaaga ttacactcag 240
gagtcccatc aaggttcagt ggcagtgggt ctggaacaga ttattctctc accattagca 300
acctggagca agaagatatt gccacttact tttgccaaca gggtaatacg cttccgtaca 360
cgttcggagg ggggaccaag ctggagatca caggtggcgg tggctcgggc ggtggtgggt 420
cgggtggcgg cggatctgag gtgaaactgc aggagtcagg acctggcctg gtggcgccct 480
cacagagcct gtccgtcaca tgcactgtct caggggtctc attacccgac tatggtgtaa 540
gctggattcg ccagcctcca cgaaagggtc tggagtggct gggagtaata tggggtagtg 600
aaaccacata ctataattca gctctcaaat ccagactgac catcatcaag gacaactcca 660
agagccaagt tttcttaaaa atgaacagtc tgcaaactga tgacacagcc atttactact 720
gtgccaaaca ttattactac ggtggtagct atgctatgga ctactggggc caaggaacct 780
cagtcaccgt ctcctcaacc acgacgccag cgccgcgacc accaacaccg gcgcccacca 840
tcgcgtcgca gcccctgtcc ctgcgcccag aggcgtgccg gccagcggcg gggggcgcag 900
tgcacacgag ggggctggac ttcgcctgtg atatctacat ctgggcgccc ttggccggga 960
cttgtggggt ccttctcctg tcactggtta tcacccttta ctgcaaacgg ggcagaaaga 1020
aactcctgta tatattcaaa caaccattta tgagaccagt acaaactact caagaggaag 1080
atggctgtag ctgccgattt ccagaagaag aagaaggagg atgtgaactg agagtgaagt 1140
tcagcaggag cgcagacgcc cccgcgtacc agcagggcca gaaccagctc tataacgagc 1200
tcaatctagg acgaagagag gagtacgatg ttttggacaa gagacgtggc cgggaccctg 1260
agatgggggg aaagccgaga aggaagaacc ctcaggaagg cctgtacaat gaactgcaga 1320
aagataagat ggcggaggcc tacagtgaga ttgggatgaa aggcgagcgc cggaggggca 1380
aggggcacga tggcctttac cagggtctca gtacagccac caaggacacc tacgacgccc 1440
ttcacatgca ggccctgccc cctcgcggaa gcggagccac caacttcagc ctgctgaagc 1500
aggccggcga cgtggaggag aaccccggcc ccatggcctt accagtgacc gccttgctcc 1560
tgccgctggc cttgctgctc cacgccgcca ggccggatat ccagatgacc cagagcccca 1620
gctccctgtc cgcatccgtg ggcgacagag tgacaattac ctgtagaagc agccaaagca 1680
tcgtgcatag cgtcggcaac acttttctgg agtggtatca acagaagccc gggaaggccc 1740
ccaaactgct gatctacaag gtgagcaaca gattcagcgg ggtcccaagc agattctccg 1800
gcagcggctc cgggactgac ttcaccctga ccattagcag cctgcagcca gaggacttcg 1860
ccacatacta ctgcttccaa gggagccagt tcccctacac cttcggccaa ggcactaagg 1920
tggagatcaa agggggggga ggaagcggcg gaggagggag cggaggcggg ggatccgaag 1980
tgcaactggt cgaatccgga ggggggctgg tccagcctgg agggtccctg agactgagct 2040
gcgccgcaag cggctacgag ttctccaggt cctggatgaa ctgggtgagg caggccccag 2100
gaaaagggct ggaatgggtg ggcaggatct accctggcga cggcgatacc aactactccg 2160
gaaagttcaa gggcaggttc actatcagcg ccgacactag caagaatacc gcctacctgc 2220
agatgaatag cctgagggcc gaggacaccg ccgtgtatta ctgcgctaga gacggcagca 2280
gctgggattg gtacttcgac gtgtggggcc agggcactct ggtgactgtg agcagcacca 2340
cgacgccagc gccgcgacca ccaacaccgg cgcccaccat cgcgtcgcag cccctgtccc 2400
tgcgcccaga ggcgtgccgg ccagcggcgg ggggcgcagt gcacacgagg gggctggact 2460
tcgcctgtga tatctacatc tgggcgccct tggccgggac ttgtggggtc cttctcctgt 2520
cactggttat caccctttac tgcaaacggg gcagaaagaa actcctgtat atattcaaac 2580
aaccatttat gagaccagta caaactactc aagaggaaga tggctgtagc tgccgatttc 2640
cagaagaaga agaaggagga tgtgaactga gagtgaagtt cagcaggagc gcagacgccc 2700
ccgcgtacca gcagggccag aaccagctct ataacgagct caatctagga cgaagagagg 2760
agtacgatgt tttggacaag agacgtggcc gggaccctga gatgggggga aagccgagaa 2820
ggaagaaccc tcaggaaggc ctgtacaatg aactgcagaa agataagatg gcggaggcct 2880
acagtgagat tgggatgaaa ggcgagcgcc ggaggggcaa ggggcacgat ggcctttacc 2940
agggtctcag tacagccacc aaggacacct acgacgccct tcacatgcag gccctgcccc 3000
ctcgctaagt ttaaac 3016
<210> 7
<211> 990
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 7
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Asp Ile Gln Met Thr Gln Thr Thr Ser Ser Leu
20 25 30
Ser Ala Ser Leu Gly Asp Arg Val Thr Ile Ser Cys Arg Ala Ser Gln
35 40 45
Asp Ile Ser Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Asp Gly Thr
50 55 60
Val Lys Leu Leu Ile Tyr His Thr Ser Arg Leu His Ser Gly Val Pro
65 70 75 80
Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Ser Leu Thr Ile
85 90 95
Ser Asn Leu Glu Gln Glu Asp Ile Ala Thr Tyr Phe Cys Gln Gln Gly
100 105 110
Asn Thr Leu Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Thr
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
130 135 140
Val Lys Leu Gln Glu Ser Gly Pro Gly Leu Val Ala Pro Ser Gln Ser
145 150 155 160
Leu Ser Val Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly
165 170 175
Val Ser Trp Ile Arg Gln Pro Pro Arg Lys Gly Leu Glu Trp Leu Gly
180 185 190
Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Asn Ser Ala Leu Lys Ser
195 200 205
Arg Leu Thr Ile Ile Lys Asp Asn Ser Lys Ser Gln Val Phe Leu Lys
210 215 220
Met Asn Ser Leu Gln Thr Asp Asp Thr Ala Ile Tyr Tyr Cys Ala Lys
225 230 235 240
His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly
245 250 255
Thr Ser Val Thr Val Ser Ser Thr Thr Thr Pro Ala Pro Arg Pro Pro
260 265 270
Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu
275 280 285
Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp
290 295 300
Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly
305 310 315 320
Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg
325 330 335
Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln
340 345 350
Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu
355 360 365
Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala
370 375 380
Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu
385 390 395 400
Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp
405 410 415
Pro Glu Met Gly Gly Lys Pro Gln Arg Arg Lys Asn Pro Gln Glu Gly
420 425 430
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
435 440 445
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
450 455 460
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
465 470 475 480
Met Gln Ala Leu Pro Pro Arg Gly Ser Gly Ala Thr Asn Phe Ser Leu
485 490 495
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ala Leu
500 505 510
Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu His Ala Ala
515 520 525
Arg Pro Asp Val Gln Ile Thr Gln Ser Pro Ser Tyr Leu Ala Ala Ser
530 535 540
Pro Gly Glu Thr Ile Thr Ile Asn Cys Arg Ala Ser Lys Ser Ile Ser
545 550 555 560
Lys Asp Leu Ala Trp Tyr Gln Glu Lys Pro Gly Lys Thr Asn Lys Leu
565 570 575
Leu Ile Tyr Ser Gly Ser Thr Leu Gln Ser Gly Ile Pro Ser Arg Phe
580 585 590
Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu
595 600 605
Glu Pro Glu Asp Phe Ala Met Tyr Tyr Cys Gln Gln His Asn Lys Tyr
610 615 620
Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly
625 630 635 640
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu
645 650 655
Gln Gln Pro Gly Ala Glu Leu Val Arg Pro Gly Ala Ser Val Lys Leu
660 665 670
Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr Trp Met Asn Trp
675 680 685
Val Lys Gln Arg Pro Asp Gln Gly Leu Glu Trp Ile Gly Arg Ile Asp
690 695 700
Pro Tyr Asp Ser Glu Thr His Tyr Asn Gln Lys Phe Lys Asp Lys Ala
705 710 715 720
Ile Leu Thr Val Asp Lys Ser Ser Ser Thr Ala Tyr Met Gln Leu Ser
725 730 735
Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys Ala Arg Gly Asn
740 745 750
Trp Asp Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val Ser Ser Thr
755 760 765
Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser
770 775 780
Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly
785 790 795 800
Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp
805 810 815
Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile
820 825 830
Thr Leu Tyr Cys Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr
835 840 845
Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln
850 855 860
Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Arg Val Lys
865 870 875 880
Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln
885 890 895
Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu
900 905 910
Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Gln Arg
915 920 925
Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys
930 935 940
Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg
945 950 955 960
Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys
965 970 975
Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
980 985 990
<210> 8
<211> 2989
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 8
gcgatcgcat ggccttacca gtgaccgcct tgctcctgcc gctggccttg ctgctccacg 60
ccgccaggcc ggacatccag atgacacaga ctacatcctc cctgtctgcc tctctgggag 120
acagagtcac catcagttgc agggcaagtc aggacattag taaatattta aattggtatc 180
agcagaaacc agatggaact gttaaactcc tgatctacca tacatcaaga ttacactcag 240
gagtcccatc aaggttcagt ggcagtgggt ctggaacaga ttattctctc accattagca 300
acctggagca agaagatatt gccacttact tttgccaaca gggtaatacg cttccgtaca 360
cgttcggagg ggggaccaag ctggagatca caggtggcgg tggctcgggc ggtggtgggt 420
cgggtggcgg cggatctgag gtgaaactgc aggagtcagg acctggcctg gtggcgccct 480
cacagagcct gtccgtcaca tgcactgtct caggggtctc attacccgac tatggtgtaa 540
gctggattcg ccagcctcca cgaaagggtc tggagtggct gggagtaata tggggtagtg 600
aaaccacata ctataattca gctctcaaat ccagactgac catcatcaag gacaactcca 660
agagccaagt tttcttaaaa atgaacagtc tgcaaactga tgacacagcc atttactact 720
gtgccaaaca ttattactac ggtggtagct atgctatgga ctactggggc caaggaacct 780
cagtcaccgt ctcctcaacc acgacgccag cgccgcgacc accaacaccg gcgcccacca 840
tcgcgtcgca gcccctgtcc ctgcgcccag aggcgtgccg gccagcggcg gggggcgcag 900
tgcacacgag ggggctggac ttcgcctgtg atatctacat ctgggcgccc ttggccggga 960
cttgtggggt ccttctcctg tcactggtta tcacccttta ctgcaaacgg ggcagaaaga 1020
aactcctgta tatattcaaa caaccattta tgagaccagt acaaactact caagaggaag 1080
atggctgtag ctgccgattt ccagaagaag aagaaggagg atgtgaactg agagtgaagt 1140
tcagcaggag cgcagacgcc cccgcgtacc agcagggcca gaaccagctc tataacgagc 1200
tcaatctagg acgaagagag gagtacgatg ttttggacaa gagacgtggc cgggaccctg 1260
agatgggggg aaagccgcag agaaggaaga accctcagga aggcctgtac aatgaactgc 1320
agaaagataa gatggcggag gcctacagtg agattgggat gaaaggcgag cgccggaggg 1380
gcaaggggca cgatggcctt taccagggtc tcagtacagc caccaaggac acctacgacg 1440
cccttcacat gcaggccctg ccccctcgcg gaagcggagc caccaacttc agcctgctga 1500
agcaggccgg cgacgtggag gagaaccccg gccccatggc cttaccagtg accgccttgc 1560
tcctgccgct ggccttgctg ctccacgccg ccaggccgga cgtgcagatc acccagagcc 1620
ccagctacct ggccgccagc cccggcgaga ccatcaccat caactgcaga gccagcaaga 1680
gcatcagcaa ggacctggcc tggtaccagg agaagcccgg caagaccaac aagctgctga 1740
tctacagcgg cagcaccctg cagagcggca tccccagcag attcagcggc agcggcagcg 1800
gcaccgactt caccctgacc atcagcagcc tggagcccga ggacttcgcc atgtactact 1860
gccagcagca caacaagtac ccctacacct tcggcggcgg caccaagctg gagatcaagg 1920
gagggggggg atccggggga ggaggctccg gcggaggcgg aagccaggtg cagctgcagc 1980
agcccggcgc cgagctggtg agacccggcg ccagcgtgaa gctgagctgc aaggccagcg 2040
gctacacctt caccagctac tggatgaact gggtgaagca gagacccgac cagggcctgg 2100
agtggatcgg cagaatcgac ccctacgaca gcgagaccca ctacaaccag aagttcaagg 2160
acaaggccat cctgaccgtg gacaagagca gcagcaccgc ctacatgcag ctgagcagcc 2220
tgaccagcga ggacagcgcc gtgtactact gcgccagagg caactgggac gactactggg 2280
gccagggcac caccctgacc gtgagcagca ccacgacgcc agcgccgcga ccaccaacac 2340
cggcgcccac catcgcgtcg cagcccctgt ccctgcgccc agaggcgtgc cggccagcgg 2400
cggggggcgc agtgcacacg agggggctgg acttcgcctg tgatatctac atctgggcgc 2460
ccttggccgg gacttgtggg gtccttctcc tgtcactggt tatcaccctt tactgcagga 2520
gtaagaggag caggctcctg cacagtgact acatgaacat gactccccgc cgccccgggc 2580
ccacccgcaa gcattaccag ccctatgccc caccacgcga cttcgcagcc tatcgctcca 2640
gagtgaagtt cagcaggagc gcagacgccc ccgcgtacca gcagggccag aaccagctct 2700
ataacgagct caatctagga cgaagagagg agtacgatgt tttggacaag agacgtggcc 2760
gggaccctga gatgggggga aagccgcaga gaaggaagaa ccctcaggaa ggcctgtaca 2820
atgaactgca gaaagataag atggcggagg cctacagtga gattgggatg aaaggcgagc 2880
gccggagggg caaggggcac gatggccttt accagggtct cagtacagcc accaaggaca 2940
cctacgacgc ccttcacatg caggccctgc cccctcgcta agtttaaac 2989
<210> 9
<211> 1066
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 9
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Ile Val Leu Thr Gln Ser Pro Ala Ile Met
20 25 30
Ser Ala Ser Pro Gly Glu Lys Val Thr Ile Thr Cys Ser Ala Ser Ser
35 40 45
Ser Ile Ser Tyr Met His Trp Phe Gln Gln Lys Pro Gly Thr Ser Pro
50 55 60
Lys Leu Trp Ile Tyr Thr Thr Ser Asn Leu Ala Ser Gly Val Pro Ala
65 70 75 80
Arg Phe Ser Gly Ser Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser
85 90 95
Arg Met Glu Ala Glu Asp Ala Ala Thr Tyr Tyr Cys His Gln Arg Ser
100 105 110
Thr Tyr Pro Leu Thr Phe Gly Ser Gly Thr Lys Leu Glu Leu Lys Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val
130 135 140
Gln Leu Gln Gln Ser Gly Ala Glu Leu Ala Lys Pro Gly Ala Ser Val
145 150 155 160
Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr Arg Met
165 170 175
His Trp Val Lys Gln Arg Pro Gly Gln Gly Leu Glu Trp Ile Gly Tyr
180 185 190
Ile Asn Pro Ser Thr Gly Tyr Thr Glu Tyr Asn Gln Lys Phe Lys Asp
195 200 205
Lys Ala Thr Leu Thr Ala Asp Lys Ser Ser Ser Thr Ala Tyr Met Gln
210 215 220
Leu Ser Ser Leu Thr Phe Glu Asp Ser Ala Val Tyr Tyr Cys Ala Arg
225 230 235 240
Gly Gly Gly Val Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val
245 250 255
Ser Ser Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr
260 265 270
Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala
275 280 285
Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile
290 295 300
Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser
305 310 315 320
Leu Val Ile Thr Leu Tyr Cys Arg Ser Lys Arg Ser Arg Leu Leu His
325 330 335
Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys
340 345 350
His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser
355 360 365
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
370 375 380
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
385 390 395 400
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
405 410 415
Ser Ala Asp Ala Pro Ala Tyr Lys Gln Gly Gln Asn Gln Leu Tyr Asn
420 425 430
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
435 440 445
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro
450 455 460
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala
465 470 475 480
Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His
485 490 495
Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp
500 505 510
Ala Leu His Met Gln Ala Leu Pro Pro Arg Gly Ser Gly Ala Thr Asn
515 520 525
Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro
530 535 540
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
545 550 555 560
His Ala Ala Arg Pro Asp Val Gln Ile Thr Gln Ser Pro Ser Tyr Leu
565 570 575
Ala Ala Ser Pro Gly Glu Thr Ile Thr Ile Asn Cys Arg Ala Ser Lys
580 585 590
Ser Ile Ser Lys Asp Leu Ala Trp Tyr Gln Glu Lys Pro Gly Lys Thr
595 600 605
Asn Lys Leu Leu Ile Tyr Ser Gly Ser Thr Leu Gln Ser Gly Ile Pro
610 615 620
Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile
625 630 635 640
Ser Ser Leu Glu Pro Glu Asp Phe Ala Met Tyr Tyr Cys Gln Gln His
645 650 655
Asn Lys Tyr Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
660 665 670
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln
675 680 685
Val Gln Leu Gln Gln Pro Gly Ala Glu Leu Val Arg Pro Gly Ala Ser
690 695 700
Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr Trp
705 710 715 720
Met Asn Trp Val Lys Gln Arg Pro Asp Gln Gly Leu Glu Trp Ile Gly
725 730 735
Arg Ile Asp Pro Tyr Asp Ser Glu Thr His Tyr Asn Gln Lys Phe Lys
740 745 750
Asp Lys Ala Ile Leu Thr Val Asp Lys Ser Ser Ser Thr Ala Tyr Met
755 760 765
Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys Ala
770 775 780
Arg Gly Asn Trp Asp Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val
785 790 795 800
Ser Ser Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr
805 810 815
Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala
820 825 830
Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile
835 840 845
Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser
850 855 860
Leu Val Ile Thr Leu Tyr Cys Arg Ser Lys Arg Ser Arg Leu Leu His
865 870 875 880
Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys
885 890 895
His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser
900 905 910
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
915 920 925
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
930 935 940
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
945 950 955 960
Ser Ala Asp Ala Pro Ala Tyr Lys Gln Gly Gln Asn Gln Leu Tyr Asn
965 970 975
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
980 985 990
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro
995 1000 1005
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu
1010 1015 1020
Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys
1025 1030 1035
Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp
1040 1045 1050
Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
1055 1060 1065
<210> 10
<211> 3217
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 10
gcgatcgcat ggccttacca gtgaccgcct tgctcctgcc gctggccttg ctgctccacg 60
ccgccaggcc gcagatcgtg ctgacccaga gccccgccat catgagcgcc agccccggcg 120
agaaggtgac catcacctgc agcgccagca gcagcatcag ctacatgcac tggttccagc 180
agaagcccgg caccagcccc aagctgtgga tctacaccac cagcaacctg gccagcggcg 240
tgcccgccag attcagcggc agcggcagcg gcaccagcta cagcctgacc atcagcagaa 300
tggaggccga ggacgccgcc acctactact gccaccagag aagcacctac cccctgacct 360
tcggcagcgg caccaagctg gagctgaagg gagggggggg atccggggga ggaggctccg 420
gcggaggcgg aagccaggtg cagctgcagc agagcggcgc cgagctggcc aagcccggcg 480
ccagcgtgaa gatgagctgc aaggccagcg gctacacctt caccagctac agaatgcact 540
gggtgaagca gagacccggc cagggcctgg agtggatcgg ctacatcaac cccagcaccg 600
gctacaccga gtacaaccag aagttcaagg acaaggccac cctgaccgcc gacaagagca 660
gcagcaccgc ctacatgcag ctgagcagcc tgaccttcga ggacagcgcc gtgtactact 720
gcgccagagg cggcggcgtg ttcgactact ggggccaggg caccaccctg accgtgagca 780
gcaccacgac gccagcgccg cgaccaccaa caccggcgcc caccatcgcg tcgcagcccc 840
tgtccctgcg cccagaggcg tgccggccag cggcgggggg cgcagtgcac acgagggggc 900
tggacttcgc ctgtgatatc tacatctggg cgcccttggc cgggacttgt ggggtccttc 960
tcctgtcact ggttatcacc ctttactgca ggagtaagag gagcaggctc ctgcacagtg 1020
actacatgaa catgactccc cgccgccccg ggcccacccg caagcattac cagccctatg 1080
ccccaccacg cgacttcgca gcctatcgct ccaaacgggg cagaaagaaa ctcctgtata 1140
tattcaaaca accatttatg agaccagtac aaactactca agaggaagat ggctgtagct 1200
gccgatttcc agaagaagaa gaaggaggat gtgaactgag agtgaagttc agcaggagcg 1260
cagacgcccc cgcgtacaag cagggccaga accagctcta taacgagctc aatctaggac 1320
gaagagagga gtacgatgtt ttggacaaga gacgtggccg ggaccctgag atggggggaa 1380
agccgagaag gaagaaccct caggaaggcc tgtacaatga actgcagaaa gataagatgg 1440
cggaggccta cagtgagatt gggatgaaag gcgagcgccg gaggggcaag gggcacgatg 1500
gcctttacca gggtctcagt acagccacca aggacaccta cgacgccctt cacatgcagg 1560
ccctgccccc tcgcggaagc ggagctacta acttcagcct gctgaagcag gctggagacg 1620
tggaggagaa ccctggacct atggccttac cagtgaccgc cttgctcctg ccgctggcct 1680
tgctgctcca cgccgccagg ccggacgtgc agatcaccca gagccccagc tacctggccg 1740
ccagccccgg cgagaccatc accatcaact gcagagccag caagagcatc agcaaggacc 1800
tggcctggta ccaggagaag cccggcaaga ccaacaagct gctgatctac agcggcagca 1860
ccctgcagag cggcatcccc agcagattca gcggcagcgg cagcggcacc gacttcaccc 1920
tgaccatcag cagcctggag cccgaggact tcgccatgta ctactgccag cagcacaaca 1980
agtaccccta caccttcggc ggcggcacca agctggagat caagggaggg gggggatccg 2040
ggggaggagg ctccggcgga ggcggaagcc aggtgcagct gcagcagccc ggcgccgagc 2100
tggtgagacc cggcgccagc gtgaagctga gctgcaaggc cagcggctac accttcacca 2160
gctactggat gaactgggtg aagcagagac ccgaccaggg cctggagtgg atcggcagaa 2220
tcgaccccta cgacagcgag acccactaca accagaagtt caaggacaag gccatcctga 2280
ccgtggacaa gagcagcagc accgcctaca tgcagctgag cagcctgacc agcgaggaca 2340
gcgccgtgta ctactgcgcc agaggcaact gggacgacta ctggggccag ggcaccaccc 2400
tgaccgtgag cagcaccacg acgccagcgc cgcgaccacc aacaccggcg cccaccatcg 2460
cgtcgcagcc cctgtccctg cgcccagagg cgtgccggcc agcggcgggg ggcgcagtgc 2520
acacgagggg gctggacttc gcctgtgata tctacatctg ggcgcccttg gccgggactt 2580
gtggggtcct tctcctgtca ctggttatca ccctttactg caggagtaag aggagcaggc 2640
tcctgcacag tgactacatg aacatgactc cccgccgccc cgggcccacc cgcaagcatt 2700
accagcccta tgccccacca cgcgacttcg cagcctatcg ctccaaacgg ggcagaaaga 2760
aactcctgta tatattcaaa caaccattta tgagaccagt acaaactact caagaggaag 2820
atggctgtag ctgccgattt ccagaagaag aagaaggagg atgtgaactg agagtgaagt 2880
tcagcaggag cgcagacgcc cccgcgtaca agcagggcca gaaccagctc tataacgagc 2940
tcaatctagg acgaagagag gagtacgatg ttttggacaa gagacgtggc cgggaccctg 3000
agatgggggg aaagccgaga aggaagaacc ctcaggaagg cctgtacaat gaactgcaga 3060
aagataagat ggcggaggcc tacagtgaga ttgggatgaa aggcgagcgc cggaggggca 3120
aggggcacga tggcctttac cagggtctca gtacagccac caaggacacc tacgacgccc 3180
ttcacatgca ggccctgccc cctcgctaag tttaaac 3217
<210> 11
<211> 985
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 11
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Ile Val Leu Thr Gln Ser Pro Ala Ile Met
20 25 30
Ser Ala Ser Pro Gly Glu Lys Val Thr Ile Thr Cys Ser Ala Ser Ser
35 40 45
Ser Ile Ser Tyr Met His Trp Phe Gln Gln Lys Pro Gly Thr Ser Pro
50 55 60
Lys Leu Trp Ile Tyr Thr Thr Ser Asn Leu Ala Ser Gly Val Pro Ala
65 70 75 80
Arg Phe Ser Gly Ser Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser
85 90 95
Arg Met Glu Ala Glu Asp Ala Ala Thr Tyr Tyr Cys His Gln Arg Ser
100 105 110
Thr Tyr Pro Leu Thr Phe Gly Ser Gly Thr Lys Leu Glu Leu Lys Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val
130 135 140
Gln Leu Gln Gln Ser Gly Ala Glu Leu Ala Lys Pro Gly Ala Ser Val
145 150 155 160
Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr Arg Met
165 170 175
His Trp Val Lys Gln Arg Pro Gly Gln Gly Leu Glu Trp Ile Gly Tyr
180 185 190
Ile Asn Pro Ser Thr Gly Tyr Thr Glu Tyr Asn Gln Lys Phe Lys Asp
195 200 205
Lys Ala Thr Leu Thr Ala Asp Lys Ser Ser Ser Thr Ala Tyr Met Gln
210 215 220
Leu Ser Ser Leu Thr Phe Glu Asp Ser Ala Val Tyr Tyr Cys Ala Arg
225 230 235 240
Gly Gly Gly Val Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val
245 250 255
Ser Ser Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr
260 265 270
Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala
275 280 285
Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile
290 295 300
Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser
305 310 315 320
Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr
325 330 335
Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu
340 345 350
Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu
355 360 365
Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln
370 375 380
Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu
385 390 395 400
Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly
405 410 415
Lys Pro Gln Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu
420 425 430
Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly
435 440 445
Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser
450 455 460
Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro
465 470 475 480
Pro Arg Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly
485 490 495
Asp Val Glu Glu Asn Pro Gly Pro Met Ala Leu Pro Val Thr Ala Leu
500 505 510
Leu Leu Pro Leu Ala Leu Leu Leu His Ala Ala Arg Pro Asp Val Gln
515 520 525
Ile Thr Gln Ser Pro Ser Tyr Leu Ala Ala Ser Pro Gly Glu Thr Ile
530 535 540
Thr Ile Asn Cys Arg Ala Ser Lys Ser Ile Ser Lys Asp Leu Ala Trp
545 550 555 560
Tyr Gln Glu Lys Pro Gly Lys Thr Asn Lys Leu Leu Ile Tyr Ser Gly
565 570 575
Ser Thr Leu Gln Ser Gly Ile Pro Ser Arg Phe Ser Gly Ser Gly Ser
580 585 590
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu Asp Phe
595 600 605
Ala Met Tyr Tyr Cys Gln Gln His Asn Lys Tyr Pro Tyr Thr Phe Gly
610 615 620
Gly Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly
625 630 635 640
Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Gln Gln Pro Gly Ala
645 650 655
Glu Leu Val Arg Pro Gly Ala Ser Val Lys Leu Ser Cys Lys Ala Ser
660 665 670
Gly Tyr Thr Phe Thr Ser Tyr Trp Met Asn Trp Val Lys Gln Arg Pro
675 680 685
Asp Gln Gly Leu Glu Trp Ile Gly Arg Ile Asp Pro Tyr Asp Ser Glu
690 695 700
Thr His Tyr Asn Gln Lys Phe Lys Asp Lys Ala Ile Leu Thr Val Asp
705 710 715 720
Lys Ser Ser Ser Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Glu
725 730 735
Asp Ser Ala Val Tyr Tyr Cys Ala Arg Gly Asn Trp Asp Asp Tyr Trp
740 745 750
Gly Gln Gly Thr Thr Leu Thr Val Ser Ser Thr Thr Thr Pro Ala Pro
755 760 765
Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu
770 775 780
Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg
785 790 795 800
Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly
805 810 815
Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Arg
820 825 830
Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr Pro
835 840 845
Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro
850 855 860
Arg Asp Phe Ala Ala Tyr Arg Ser Arg Val Lys Phe Ser Arg Ser Ala
865 870 875 880
Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu
885 890 895
Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly
900 905 910
Arg Asp Pro Glu Met Gly Gly Lys Pro Gln Arg Arg Lys Asn Pro Gln
915 920 925
Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr
930 935 940
Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp
945 950 955 960
Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala
965 970 975
Leu His Met Gln Ala Leu Pro Pro Arg
980 985
<210> 12
<211> 2974
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 12
gcgatcgcat ggccttacca gtgaccgcct tgctcctgcc gctggccttg ctgctccacg 60
ccgccaggcc gcagatcgtg ctgacccaga gccccgccat catgagcgcc agccccggcg 120
agaaggtgac catcacctgc agcgccagca gcagcatcag ctacatgcac tggttccagc 180
agaagcccgg caccagcccc aagctgtgga tctacaccac cagcaacctg gccagcggcg 240
tgcccgccag attcagcggc agcggcagcg gcaccagcta cagcctgacc atcagcagaa 300
tggaggccga ggacgccgcc acctactact gccaccagag aagcacctac cccctgacct 360
tcggcagcgg caccaagctg gagctgaagg gagggggggg atccggggga ggaggctccg 420
gcggaggcgg aagccaggtg cagctgcagc agagcggcgc cgagctggcc aagcccggcg 480
ccagcgtgaa gatgagctgc aaggccagcg gctacacctt caccagctac agaatgcact 540
gggtgaagca gagacccggc cagggcctgg agtggatcgg ctacatcaac cccagcaccg 600
gctacaccga gtacaaccag aagttcaagg acaaggccac cctgaccgcc gacaagagca 660
gcagcaccgc ctacatgcag ctgagcagcc tgaccttcga ggacagcgcc gtgtactact 720
gcgccagagg cggcggcgtg ttcgactact ggggccaggg caccaccctg accgtgagca 780
gcaccacgac gccagcgccg cgaccaccaa caccggcgcc caccatcgcg tcgcagcccc 840
tgtccctgcg cccagaggcg tgccggccag cggcgggggg cgcagtgcac acgagggggc 900
tggacttcgc ctgtgatatc tacatctggg cgcccttggc cgggacttgt ggggtccttc 960
tcctgtcact ggttatcacc ctttactgca aacggggcag aaagaaactc ctgtatatat 1020
tcaaacaacc atttatgaga ccagtacaaa ctactcaaga ggaagatggc tgtagctgcc 1080
gatttccaga agaagaagaa ggaggatgtg aactgagagt gaagttcagc aggagcgcag 1140
acgcccccgc gtaccagcag ggccagaacc agctctataa cgagctcaat ctaggacgaa 1200
gagaggagta cgatgttttg gacaagagac gtggccggga ccctgagatg gggggaaagc 1260
cgcagagaag gaagaaccct caggaaggcc tgtacaatga actgcagaaa gataagatgg 1320
cggaggccta cagtgagatt gggatgaaag gcgagcgccg gaggggcaag gggcacgatg 1380
gcctttacca gggtctcagt acagccacca aggacaccta cgacgccctt cacatgcagg 1440
ccctgccccc tcgcggaagc ggagctacta acttcagcct gctgaagcag gctggagacg 1500
tggaggagaa ccctggacct atggccttac cagtgaccgc cttgctcctg ccgctggcct 1560
tgctgctcca cgccgccagg ccggacgtgc agatcaccca gagccccagc tacctggccg 1620
ccagccccgg cgagaccatc accatcaact gcagagccag caagagcatc agcaaggacc 1680
tggcctggta ccaggagaag cccggcaaga ccaacaagct gctgatctac agcggcagca 1740
ccctgcagag cggcatcccc agcagattca gcggcagcgg cagcggcacc gacttcaccc 1800
tgaccatcag cagcctggag cccgaggact tcgccatgta ctactgccag cagcacaaca 1860
agtaccccta caccttcggc ggcggcacca agctggagat caagggaggg gggggatccg 1920
ggggaggagg ctccggcgga ggcggaagcc aggtgcagct gcagcagccc ggcgccgagc 1980
tggtgagacc cggcgccagc gtgaagctga gctgcaaggc cagcggctac accttcacca 2040
gctactggat gaactgggtg aagcagagac ccgaccaggg cctggagtgg atcggcagaa 2100
tcgaccccta cgacagcgag acccactaca accagaagtt caaggacaag gccatcctga 2160
ccgtggacaa gagcagcagc accgcctaca tgcagctgag cagcctgacc agcgaggaca 2220
gcgccgtgta ctactgcgcc agaggcaact gggacgacta ctggggccag ggcaccaccc 2280
tgaccgtgag cagcaccacg acgccagcgc cgcgaccacc aacaccggcg cccaccatcg 2340
cgtcgcagcc cctgtccctg cgcccagagg cgtgccggcc agcggcgggg ggcgcagtgc 2400
acacgagggg gctggacttc gcctgtgata tctacatctg ggcgcccttg gccgggactt 2460
gtggggtcct tctcctgtca ctggttatca ccctttactg caggagtaag aggagcaggc 2520
tcctgcacag tgactacatg aacatgactc cccgccgccc cgggcccacc cgcaagcatt 2580
accagcccta tgccccacca cgcgacttcg cagcctatcg ctccagagtg aagttcagca 2640
ggagcgcaga cgcccccgcg taccagcagg gccagaacca gctctataac gagctcaatc 2700
taggacgaag agaggagtac gatgttttgg acaagagacg tggccgggac cctgagatgg 2760
ggggaaagcc gcagagaagg aagaaccctc aggaaggcct gtacaatgaa ctgcagaaag 2820
ataagatggc ggaggcctac agtgagattg ggatgaaagg cgagcgccgg aggggcaagg 2880
ggcacgatgg cctttaccag ggtctcagta cagccaccaa ggacacctac gacgcccttc 2940
acatgcaggc cctgccccct cgctaagttt aaac 2974
<210> 13
<211> 546
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 13
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Ser Asp Ile Val Leu Thr Gln Ser Pro Ala Ser
20 25 30
Leu Ala Val Ser Leu Gly Gln Arg Ala Thr Ile Ser Cys Arg Ala Ser
35 40 45
Lys Ser Val Ser Thr Ser Gly Tyr Ser Tyr Leu His Trp Tyr Gln Gln
50 55 60
Lys Pro Gly Gln Pro Pro Lys Leu Leu Ile Tyr Leu Ala Ser Asn Leu
65 70 75 80
Glu Ser Gly Val Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
85 90 95
Phe Thr Leu Asn Ile His Pro Val Glu Glu Glu Asp Ala Ala Thr Tyr
100 105 110
Tyr Cys Gln His Ser Arg Glu Leu Pro Phe Thr Phe Gly Ser Gly Thr
115 120 125
Lys Leu Glu Ile Lys Lys Ile Ser Gly Gly Gly Gly Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
145 150 155 160
Ser Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
165 170 175
Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe Ser Arg
180 185 190
Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
195 200 205
Ile Gly Glu Ile Asn Pro Thr Ser Ser Thr Ile Asn Phe Thr Pro Ser
210 215 220
Leu Lys Asp Lys Val Phe Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu
225 230 235 240
Tyr Leu Gln Met Ser Lys Val Arg Ser Glu Asp Thr Ala Leu Tyr Tyr
245 250 255
Cys Ala Arg Gly Asn Tyr Tyr Arg Tyr Gly Asp Ala Met Asp Tyr Trp
260 265 270
Gly Gln Gly Thr Ser Val Thr Val Ser Thr Thr Thr Pro Ala Pro Arg
275 280 285
Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg
290 295 300
Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly
305 310 315 320
Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr
325 330 335
Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Arg Ser
340 345 350
Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr Pro Arg
355 360 365
Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg
370 375 380
Asp Phe Ala Ala Tyr Arg Ser Lys Arg Gly Arg Lys Lys Leu Leu Tyr
385 390 395 400
Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu
405 410 415
Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu
420 425 430
Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln
435 440 445
Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu
450 455 460
Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly
465 470 475 480
Lys Pro Gln Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu
485 490 495
Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly
500 505 510
Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser
515 520 525
Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro
530 535 540
Pro Arg
545
<210> 14
<211> 1657
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 14
gcgatcgcat ggccttacca gtgaccgcct tgctcctgcc gctggccttg ctgctccacg 60
ccgccaggcc gagcgacatc gtgctgaccc agagccccgc cagcctggcc gtgagcctgg 120
gccagagagc caccatcagc tgcagagcca gcaagagcgt gagcaccagc ggctacagct 180
acctgcactg gtaccagcag aagcccggcc agccccccaa gctgctgatc tacctggcca 240
gcaacctgga gagcggcgtg cccgccagat tcagcggcag cggcagcggc accgacttca 300
ccctgaacat ccaccccgtg gaggaggagg acgccgccac ctactactgc cagcacagca 360
gagagctgcc cttcaccttc ggcagcggca ccaagctgga gatcaagaag atcagcggcg 420
gcggcggcag cggcggcggc ggcagcggcg gcggcggcag cggcggcggc ggcagcggcg 480
gcggcggcag ccaggtgcag ctggtggaga gcggcggcgg cctggtgcag cccggcggca 540
gcctgaagct gagctgcgcc gccagcggct tcgacttcag cagatactgg atgagctggg 600
tgagacaggc ccccggcaag ggcctggagt ggatcggcga gatcaacccc accagcagca 660
ccatcaactt cacccccagc ctgaaggaca aggtgttcat cagcagagac aacgccaaga 720
acaccctgta cctgcagatg agcaaggtga gaagcgagga caccgccctg tactactgcg 780
ccagaggcaa ctactacaga tacggcgacg ccatggacta ctggggccag ggcaccagcg 840
tgaccgtgag caccacgacg ccagcgccgc gaccaccaac accggcgccc accatcgcgt 900
cgcagcccct gtccctgcgc ccagaggcgt gccggccagc ggcggggggc gcagtgcaca 960
cgagggggct ggacttcgcc tgtgatatct acatctgggc gcccttggcc gggacttgtg 1020
gggtccttct cctgtcactg gttatcaccc tttactgcag gagtaagagg agcaggctcc 1080
tgcacagtga ctacatgaac atgactcccc gccgccccgg gcccacccgc aagcattacc 1140
agccctatgc cccaccacgc gacttcgcag cctatcgctc caaacggggc agaaagaaac 1200
tcctgtatat attcaaacaa ccatttatga gaccagtaca aactactcaa gaggaagatg 1260
gctgtagctg ccgatttcca gaagaagaag aaggaggatg tgaactgaga gtgaagttca 1320
gcaggagcgc agacgccccc gcgtaccagc agggccagaa ccagctctat aacgagctca 1380
atctaggacg aagagaggag tacgatgttt tggacaagag acgtggccgg gaccctgaga 1440
tggggggaaa gccgcagaga aggaagaacc ctcaggaagg cctgtacaat gaactgcaga 1500
aagataagat ggcggaggcc tacagtgaga ttgggatgaa aggcgagcgc cggaggggca 1560
aggggcacga tggcctttac cagggtctca gtacagccac caaggacacc tacgacgccc 1620
ttcacatgca ggccctgccc cctcgctaag tttaaac 1657
<210> 15
<211> 490
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 15
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Asp Ile Val Leu Thr Gln Ser Pro Ala Ser Leu
20 25 30
Ala Val Ser Leu Gly Gln Arg Ala Thr Ile Ser Cys Arg Ala Ser Lys
35 40 45
Ser Val Ser Thr Ser Gly Tyr Ser Tyr Leu His Trp Tyr Gln Gln Lys
50 55 60
Pro Gly Gln Pro Pro Lys Leu Leu Ile Tyr Leu Ala Ser Asn Leu Glu
65 70 75 80
Ser Gly Val Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
85 90 95
Thr Leu Asn Ile His Pro Val Glu Glu Glu Asp Ala Ala Thr Tyr Tyr
100 105 110
Cys Gln His Ser Arg Glu Leu Pro Phe Thr Phe Gly Ser Gly Thr Lys
115 120 125
Leu Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
130 135 140
Gly Gly Ser Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
145 150 155 160
Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe
165 170 175
Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
180 185 190
Glu Trp Ile Gly Glu Ile Asn Pro Thr Ser Ser Thr Ile Asn Phe Thr
195 200 205
Pro Ser Leu Lys Asp Lys Val Phe Ile Ser Arg Asp Asn Ala Lys Asn
210 215 220
Thr Leu Tyr Leu Gln Met Ser Lys Val Arg Ser Glu Asp Thr Ala Leu
225 230 235 240
Tyr Tyr Cys Ala Arg Gly Asn Tyr Tyr Arg Tyr Gly Asp Ala Met Asp
245 250 255
Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Thr Thr Thr Pro Ala
260 265 270
Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser
275 280 285
Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr
290 295 300
Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala
305 310 315 320
Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys
325 330 335
Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr
340 345 350
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
355 360 365
Pro Arg Asp Phe Ala Ala Tyr Arg Ser Arg Val Lys Phe Ser Arg Ser
370 375 380
Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu
385 390 395 400
Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg
405 410 415
Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Gln Arg Arg Lys Asn Pro
420 425 430
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala
435 440 445
Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His
450 455 460
Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp
465 470 475 480
Ala Leu His Met Gln Ala Leu Pro Pro Arg
485 490
<210> 16
<211> 1489
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 16
gcgatcgcat ggccttacca gtgaccgcct tgctcctgcc gctggccttg ctgctccacg 60
ccgccaggcc ggacatcgtg ctgacccaga gccccgccag cctggccgtg agcctgggcc 120
agagggccac catcagctgc agggccagca agagcgtgag caccagcggc tacagctacc 180
tgcactggta ccagcagaag cccggccagc cccccaagct gctgatctac ctggccagca 240
acctggagag cggcgtgccc gccaggttca gcggcagcgg cagcggcacc gacttcaccc 300
tgaacatcca ccccgtggag gaggaggacg ccgccaccta ctactgccag cacagcaggg 360
agctgccctt caccttcggc agcggcacca agctggagat caagggaggg gggggatccg 420
ggggaggagg ctccggcgga ggcggaagcc aggtgcagct ggtggagagc ggcggcggcc 480
tggtgcagcc cggcggcagc ctgaagctga gctgcgccgc cagcggcttc gacttcagca 540
ggtactggat gagctgggtg aggcaggccc ccggcaaggg cctggagtgg atcggcgaga 600
tcaaccccac cagcagcacc atcaacttca cccccagcct gaaggacaag gtgttcatca 660
gcagggacaa cgccaagaac accctgtacc tgcagatgag caaggtgagg agcgaggaca 720
ccgccctgta ctactgcgcc aggggcaact actacaggta cggcgacgcc atggactact 780
ggggccaggg caccagcgtg accgtgagca ccacgacgcc agcgccgcga ccaccaacac 840
cggcgcccac catcgcgtcg cagcccctgt ccctgcgccc agaggcgtgc cggccagcgg 900
cggggggcgc agtgcacacg agggggctgg acttcgcctg tgatatctac atctgggcgc 960
ccttggccgg gacttgtggg gtccttctcc tgtcactggt tatcaccctt tactgcagga 1020
gtaagaggag caggctcctg cacagtgact acatgaacat gactccccgc cgccccgggc 1080
ccacccgcaa gcattaccag ccctatgccc caccacgcga cttcgcagcc tatcgctcca 1140
gagtgaagtt cagcaggagc gcagacgccc ccgcgtacca gcagggccag aaccagctct 1200
ataacgagct caatctagga cgaagagagg agtacgatgt tttggacaag agacgtggcc 1260
gggaccctga gatgggggga aagccgcaga gaaggaagaa ccctcaggaa ggcctgtaca 1320
atgaactgca gaaagataag atggcggagg cctacagtga gattgggatg aaaggcgagc 1380
gccggagggg caaggggcac gatggccttt accagggtct cagtacagcc accaaggaca 1440
cctacgacgc ccttcacatg caggccctgc cccctcgcta agtttaaac 1489
<210> 17
<211> 491
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 17
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Asp Ile Val Leu Thr Gln Ser Pro Ala Ser Leu
20 25 30
Ala Val Ser Leu Gly Gln Arg Ala Thr Ile Ser Cys Arg Ala Ser Lys
35 40 45
Ser Val Ser Thr Ser Gly Tyr Ser Tyr Leu His Trp Tyr Gln Gln Lys
50 55 60
Pro Gly Gln Pro Pro Lys Leu Leu Ile Tyr Leu Ala Ser Asn Leu Glu
65 70 75 80
Ser Gly Val Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
85 90 95
Thr Leu Asn Ile His Pro Val Glu Glu Glu Asp Ala Ala Thr Tyr Tyr
100 105 110
Cys Gln His Ser Arg Glu Leu Pro Phe Thr Phe Gly Ser Gly Thr Lys
115 120 125
Leu Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
130 135 140
Gly Gly Ser Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
145 150 155 160
Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe
165 170 175
Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
180 185 190
Glu Trp Ile Gly Glu Ile Asn Pro Thr Ser Ser Thr Ile Asn Phe Thr
195 200 205
Pro Ser Leu Lys Asp Lys Val Phe Ile Ser Arg Asp Asn Ala Lys Asn
210 215 220
Thr Leu Tyr Leu Gln Met Ser Lys Val Arg Ser Glu Asp Thr Ala Leu
225 230 235 240
Tyr Tyr Cys Ala Arg Gly Asn Tyr Tyr Arg Tyr Gly Asp Ala Met Asp
245 250 255
Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Thr Thr Thr Pro Ala
260 265 270
Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser
275 280 285
Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr
290 295 300
Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala
305 310 315 320
Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys
325 330 335
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
340 345 350
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
355 360 365
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
370 375 380
Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn
385 390 395 400
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
405 410 415
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Gln Arg Arg Lys Asn
420 425 430
Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu
435 440 445
Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly
450 455 460
His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr
465 470 475 480
Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
485 490
<210> 18
<211> 1492
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 18
gcgatcgcat ggccttacca gtgaccgcct tgctcctgcc gctggccttg ctgctccacg 60
ccgccaggcc ggacatcgtg ctgacccaga gccccgccag cctggccgtg agcctgggcc 120
agagggccac catcagctgc agggccagca agagcgtgag caccagcggc tacagctacc 180
tgcactggta ccagcagaag cccggccagc cccccaagct gctgatctac ctggccagca 240
acctggagag cggcgtgccc gccaggttca gcggcagcgg cagcggcacc gacttcaccc 300
tgaacatcca ccccgtggag gaggaggacg ccgccaccta ctactgccag cacagcaggg 360
agctgccctt caccttcggc agcggcacca agctggagat caagggaggg gggggatccg 420
ggggaggagg ctccggcgga ggcggaagcc aggtgcagct ggtggagagc ggcggcggcc 480
tggtgcagcc cggcggcagc ctgaagctga gctgcgccgc cagcggcttc gacttcagca 540
ggtactggat gagctgggtg aggcaggccc ccggcaaggg cctggagtgg atcggcgaga 600
tcaaccccac cagcagcacc atcaacttca cccccagcct gaaggacaag gtgttcatca 660
gcagggacaa cgccaagaac accctgtacc tgcagatgag caaggtgagg agcgaggaca 720
ccgccctgta ctactgcgcc aggggcaact actacaggta cggcgacgcc atggactact 780
ggggccaggg caccagcgtg accgtgagca ccacgacgcc agcgccgcga ccaccaacac 840
cggcgcccac catcgcgtcg cagcccctgt ccctgcgccc agaggcgtgc cggccagcgg 900
cggggggcgc agtgcacacg agggggctgg acttcgcctg tgatatctac atctgggcgc 960
ccttggccgg gacttgtggg gtccttctcc tgtcactggt tatcaccctt tactgcaaac 1020
ggggcagaaa gaaactcctg tatatattca aacaaccatt tatgagacca gtacaaacta 1080
ctcaagagga agatggctgt agctgccgat ttccagaaga agaagaagga ggatgtgaac 1140
tgagagtgaa gttcagcagg agcgcagacg cccccgcgta ccagcagggc cagaaccagc 1200
tctataacga gctcaatcta ggacgaagag aggagtacga tgttttggac aagagacgtg 1260
gccgggaccc tgagatgggg ggaaagccgc agagaaggaa gaaccctcag gaaggcctgt 1320
acaatgaact gcagaaagat aagatggcgg aggcctacag tgagattggg atgaaaggcg 1380
agcgccggag gggcaagggg cacgatggcc tttaccaggg tctcagtaca gccaccaagg 1440
acacctacga cgcccttcac atgcaggccc tgccccctcg ctaagtttaa ac 1492
<210> 19
<211> 997
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 19
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Asp Val Val Met Thr Gln Ser His Arg Phe Met
20 25 30
Ser Thr Ser Val Gly Asp Arg Val Ser Ile Thr Cys Arg Ala Ser Gln
35 40 45
Asp Val Asn Thr Ala Val Ser Trp Tyr Gln Gln Lys Pro Gly Gln Ser
50 55 60
Pro Lys Leu Leu Ile Phe Ser Ala Ser Tyr Arg Tyr Thr Gly Val Pro
65 70 75 80
Asp Arg Phe Thr Gly Ser Gly Ser Gly Ala Asp Phe Thr Leu Thr Ile
85 90 95
Ser Ser Val Gln Ala Glu Asp Leu Ala Val Tyr Tyr Cys Gln Gln His
100 105 110
Tyr Ser Thr Pro Trp Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln
130 135 140
Ile Gln Leu Val Gln Ser Gly Pro Asp Leu Lys Lys Pro Gly Glu Thr
145 150 155 160
Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asn Phe Gly
165 170 175
Met Asn Trp Val Lys Gln Ala Pro Gly Lys Gly Phe Lys Trp Met Ala
180 185 190
Trp Ile Asn Thr Thr Arg Tyr Thr Gly Glu Ser Tyr Phe Ala Asp Asp
195 200 205
Phe Lys Gly Arg Phe Ala Phe Ser Val Glu Thr Ser Ala Thr Thr Ala
210 215 220
Tyr Leu Gln Ile Asn Asn Leu Lys Thr Glu Asp Thr Ala Thr Tyr Phe
225 230 235 240
Cys Ala Arg Gly Glu Ile Tyr Tyr Gly Tyr Asp Gly Gly Phe Ala Tyr
245 250 255
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ala Thr Thr Thr Pro Ala
260 265 270
Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser
275 280 285
Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr
290 295 300
Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala
305 310 315 320
Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys
325 330 335
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
340 345 350
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
355 360 365
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
370 375 380
Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn
385 390 395 400
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
405 410 415
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Gln Arg Arg Lys Asn
420 425 430
Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu
435 440 445
Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly
450 455 460
His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr
465 470 475 480
Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg Gly Ser Gly Ala Thr
485 490 495
Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly
500 505 510
Pro Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
515 520 525
His Ala Ala Arg Pro Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu
530 535 540
Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln
545 550 555 560
Asp Val Gly Ile Ala Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val
565 570 575
Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg His Thr Gly Val Pro
580 585 590
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile
595 600 605
Ser Ser Leu Gln Pro Glu Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr
610 615 620
Ser Ser Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
625 630 635 640
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
645 650 655
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
660 665 670
Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe Ser Arg Tyr Trp
675 680 685
Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile Gly
690 695 700
Glu Ile Asn Pro Asp Ser Ser Thr Ile Asn Tyr Ala Pro Ser Leu Lys
705 710 715 720
Asp Lys Phe Ile Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu
725 730 735
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
740 745 750
Arg Pro Asp Gly Asn Tyr Trp Tyr Phe Asp Val Trp Gly Gln Gly Thr
755 760 765
Leu Val Thr Val Ser Ser Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr
770 775 780
Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala
785 790 795 800
Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe
805 810 815
Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val
820 825 830
Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Arg Ser Lys Arg Ser
835 840 845
Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly
850 855 860
Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala
865 870 875 880
Ala Tyr Arg Ser Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala
885 890 895
Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg
900 905 910
Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu
915 920 925
Met Gly Gly Lys Pro Gln Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr
930 935 940
Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly
945 950 955 960
Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln
965 970 975
Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln
980 985 990
Ala Leu Pro Pro Arg
995
<210> 20
<211> 2994
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 20
atggccttac cagtgaccgc cttgctcctg ccgctggcct tgctgctcca cgccgccagg 60
ccggatgtgg tgatgaccca gagccatcgc tttatgagca ccagcgtggg cgatcgcgtg 120
agcattacct gccgcgcgag ccaggatgtg aacaccgcgg tgagctggta tcagcagaaa 180
ccgggccaga gcccgaaact gctgattttt agcgcgagct atcgctatac cggcgtgccg 240
gatcgcttta ccggcagcgg cagcggcgcg gattttaccc tgaccattag cagcgtgcag 300
gcggaagatc tggcggtgta ttattgccag cagcattata gcaccccgtg gacctttggc 360
ggcggcacca aactggaaat taaaggaggg gggggatccg ggggaggagg ctccggcgga 420
ggcggaagcc agattcagct ggtgcagagc ggcccggatc tgaaaaaacc gggcgaaacc 480
gtgaaactga gctgcaaagc gagcggctat acctttacca actttggcat gaactgggtg 540
aaacaggcgc cgggcaaagg ctttaaatgg atggcgtgga ttaacaccac ccgctatacc 600
ggcgaaagct attttgcgga tgattttaaa ggccgctttg cgtttagcgt ggaaaccagc 660
gcgaccaccg cgtatctgca gattaacaac ctgaaaaccg aagataccgc gacctatttt 720
tgcgcgcgcg gcgaaattta ttatggctat gatggcggct ttgcgtattg gggccagggc 780
accctggtga ccgtgagcgc gaccacgacg ccagcgccgc gaccaccaac accggcgccc 840
accatcgcgt cgcagcccct gtccctgcgc ccagaggcgt gccggccagc ggcggggggc 900
gcagtgcaca cgagggggct ggacttcgcc tgtgatatct acatctgggc gcccttggcc 960
gggacttgtg gggtccttct cctgtcactg gttatcaccc tttactgcaa acggggcaga 1020
aagaaactcc tgtatatatt caaacaacca tttatgagac cagtacaaac tactcaagag 1080
gaagatggct gtagctgccg atttccagaa gaagaagaag gaggatgtga actgagagtg 1140
aagttcagca ggagcgcaga cgcccccgcg taccagcagg gccagaacca gctctataac 1200
gagctcaatc taggacgaag agaggagtac gatgttttgg acaagagacg tggccgggac 1260
cctgagatgg ggggaaagcc gcagagaagg aagaaccctc aggaaggcct gtacaatgaa 1320
ctgcagaaag ataagatggc ggaggcctac agtgagattg ggatgaaagg cgagcgccgg 1380
aggggcaagg ggcacgatgg cctttaccag ggtctcagta cagccaccaa ggacacctac 1440
gacgcccttc acatgcaggc cctgccccct cgcggaagcg gagccaccaa cttcagcctg 1500
ctgaagcagg ccggcgacgt ggaggagaac cccggccccg ccttaccagt gaccgccttg 1560
ctcctgccgc tggccttgct gctccacgcc gccaggccgg atattcagat gacccagagc 1620
ccgagcagcc tgagcgcgag cgtgggcgat cgcgtgacca ttacctgcaa agcgagccag 1680
gatgtgggca ttgcggtggc gtggtatcag cagaaaccgg gcaaagtgcc gaaactgctg 1740
atttattggg cgagcacccg ccataccggc gtgccggatc gctttagcgg cagcggcagc 1800
ggcaccgatt ttaccctgac cattagcagc ctgcagccgg aagatgtggc gacctattat 1860
tgccagcagt atagcagcta tccgtatacc tttggccagg gcaccaaagt ggaaattaaa 1920
ggaggggggg gatccggggg aggaggctcc ggcggaggcg gaagcgaagt gcagctggtg 1980
gaaagcggcg gcggcctggt gcagccgggc ggcagcctgc gcctgagctg cgcggcgagc 2040
ggctttgatt ttagccgcta ttggatgagc tgggtgcgcc aggcgccggg caaaggcctg 2100
gaatggattg gcgaaattaa cccggatagc agcaccatta actatgcgcc gagcctgaaa 2160
gataaattta ttattagccg cgataacgcg aaaaacagcc tgtatctgca gatgaacagc 2220
ctgcgcgcgg aagataccgc ggtgtattat tgcgcgcgcc cggatggcaa ctattggtat 2280
tttgatgtgt ggggccaggg caccctggtg accgtgagca gcaccacgac gccagcgccg 2340
cgaccaccaa caccggcgcc caccatcgcg tcgcagcccc tgtccctgcg cccagaggcg 2400
tgccggccag cggcgggggg cgcagtgcac acgagggggc tggacttcgc ctgtgatatc 2460
tacatctggg cgcccttggc cgggacttgt ggggtccttc tcctgtcact ggttatcacc 2520
ctttactgca ggagtaagag gagcaggctc ctgcacagtg actacatgaa catgactccc 2580
cgccgccccg ggcccacccg caagcattac cagccctatg ccccaccacg cgacttcgca 2640
gcctatcgct ccagagtgaa gttcagcagg agcgcagacg cccccgcgta ccagcagggc 2700
cagaaccagc tctataacga gctcaatcta ggacgaagag aggagtacga tgttttggac 2760
aagagacgtg gccgggaccc tgagatgggg ggaaagccgc agagaaggaa gaaccctcag 2820
gaaggcctgt acaatgaact gcagaaagat aagatggcgg aggcctacag tgagattggg 2880
atgaaaggcg agcgccggag gggcaagggg cacgatggcc tttaccaggg tctcagtaca 2940
gccaccaagg acacctacga cgcccttcac atgcaggccc tgccccctcg ctaa 2994
<210> 21
<211> 997
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 21
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Asp Val Val Met Thr Gln Ser His Arg Phe Met
20 25 30
Ser Thr Ser Val Gly Asp Arg Val Ser Ile Thr Cys Arg Ala Ser Gln
35 40 45
Asp Val Asn Thr Ala Val Ser Trp Tyr Gln Gln Lys Pro Gly Gln Ser
50 55 60
Pro Lys Leu Leu Ile Phe Ser Ala Ser Tyr Arg Tyr Thr Gly Val Pro
65 70 75 80
Asp Arg Phe Thr Gly Ser Gly Ser Gly Ala Asp Phe Thr Leu Thr Ile
85 90 95
Ser Ser Val Gln Ala Glu Asp Leu Ala Val Tyr Tyr Cys Gln Gln His
100 105 110
Tyr Ser Thr Pro Trp Thr Phe Gly Gly Gly Thr Lys Leu Asp Ile Lys
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln
130 135 140
Ile Gln Leu Val Gln Ser Gly Pro Asp Leu Lys Lys Pro Gly Glu Thr
145 150 155 160
Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asn Phe Gly
165 170 175
Met Asn Trp Val Lys Gln Ala Pro Gly Lys Gly Phe Lys Trp Met Ala
180 185 190
Trp Ile Asn Thr Tyr Thr Gly Glu Ser Tyr Phe Ala Asp Asp Phe Lys
195 200 205
Gly Arg Phe Ala Phe Ser Val Glu Thr Ser Ala Thr Thr Ala Tyr Leu
210 215 220
Gln Ile Asn Asn Leu Lys Thr Glu Asp Thr Ala Thr Tyr Phe Cys Ala
225 230 235 240
Arg Gly Glu Ile Tyr Tyr Gly Tyr Asp Gly Gly Phe Ala Tyr Trp Gly
245 250 255
Gln Gly Thr Leu Val Thr Val Ser Ala Thr Thr Thr Pro Ala Pro Arg
260 265 270
Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg
275 280 285
Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly
290 295 300
Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr
305 310 315 320
Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg
325 330 335
Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro
340 345 350
Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu
355 360 365
Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala
370 375 380
Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu
385 390 395 400
Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly
405 410 415
Arg Asp Pro Glu Met Gly Gly Lys Pro Gln Arg Arg Lys Asn Pro Gln
420 425 430
Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr
435 440 445
Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp
450 455 460
Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala
465 470 475 480
Leu His Met Gln Ala Leu Pro Pro Arg Gly Ser Gly Ala Thr Asn Phe
485 490 495
Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met
500 505 510
Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu His
515 520 525
Ala Ala Arg Pro Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
530 535 540
Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp
545 550 555 560
Val Gly Ile Ala Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro
565 570 575
Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp
580 585 590
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
595 600 605
Ser Leu Gln Pro Glu Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser
610 615 620
Ser Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Gly
625 630 635 640
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val
645 650 655
Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu
660 665 670
Arg Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe Ser Arg Tyr Trp Met
675 680 685
Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile Gly Glu
690 695 700
Ile Asn Pro Asp Ser Ser Thr Ile Asn Tyr Ala Pro Ser Leu Lys Asp
705 710 715 720
Lys Phe Ile Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln
725 730 735
Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg
740 745 750
Pro Asp Gly Asn Tyr Trp Tyr Phe Asp Val Trp Gly Gln Gly Thr Leu
755 760 765
Val Thr Val Ser Ser Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro
770 775 780
Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys
785 790 795 800
Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala
805 810 815
Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu
820 825 830
Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys
835 840 845
Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr
850 855 860
Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly
865 870 875 880
Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala
885 890 895
Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg
900 905 910
Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu
915 920 925
Met Gly Gly Lys Pro Gln Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr
930 935 940
Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly
945 950 955 960
Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln
965 970 975
Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln
980 985 990
Ala Leu Pro Pro Arg
995
<210> 22
<211> 3009
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 22
cgatcgcatg gccttaccag tgaccgcctt gctcctgccg ctggccttgc tgctccacgc 60
cgccaggccg gatgtggtga tgacccagag ccatcgcttt atgagcacca gcgtgggaga 120
tcgagtgagc attacctgcc gcgcgagcca ggatgtgaac accgcggtga gctggtatca 180
gcagaaaccg ggccagagcc cgaaactgct gatttttagc gcgagctatc gctataccgg 240
cgtgccggat cgctttaccg gcagcggcag cggcgcggat tttaccctga ccattagcag 300
cgtgcaggcg gaagatctgg cggtgtatta ttgccagcag cattatagca ccccgtggac 360
ctttggcggc ggcaccaaac tggatattaa aggagggggg ggatccgggg gaggaggctc 420
cggcggaggc ggaagccaga ttcagctggt gcagagcggc ccggatctga aaaaaccggg 480
cgaaaccgtg aaactgagct gcaaagcgag cggctatacc tttaccaact ttggcatgaa 540
ctgggtgaaa caggcgccgg gcaaaggctt taaatggatg gcgtggatta acacctatac 600
cggcgaaagc tattttgcgg atgattttaa aggccgcttt gcgtttagcg tggaaaccag 660
cgcgaccacc gcgtatctgc agattaacaa cctgaaaacc gaagataccg cgacctattt 720
ttgcgcgcgc ggcgaaattt attatggcta tgatggcggc tttgcgtatt ggggccaggg 780
caccctggtg accgtgagcg cgaccacgac gccagcgccg cgaccaccaa caccggcgcc 840
caccatcgcg tcgcagcccc tgtccctgcg cccagaggcg tgccggccag cggcgggggg 900
cgcagtgcac acgagggggc tggacttcgc ctgtgatatc tacatctggg cgcccttggc 960
cgggacttgt ggggtccttc tcctgtcact ggttatcacc ctttactgca aacggggcag 1020
aaagaaactc ctgtatatat tcaagcaacc atttatgaga ccagtacaaa ctactcaaga 1080
ggaagatggc tgtagctgcc gatttccaga agaagaagaa ggaggatgtg aactgagagt 1140
gaagttcagc aggagcgcag acgcccccgc gtaccagcag ggccagaacc agctctataa 1200
cgagctcaat ctaggacgaa gagaggagta cgatgttttg gacaagagac gtggccggga 1260
ccctgagatg gggggaaagc cgcagagaag gaagaaccct caggaaggcc tgtacaatga 1320
actgcagaaa gataagatgg cggaggccta cagtgagatt gggatgaaag gcgagcgccg 1380
gaggggcaag gggcacgatg gcctttacca gggtctcagt acagccacca aggacaccta 1440
cgacgccctt cacatgcagg ccctgccccc tcgcggaagc ggagccacca acttcagcct 1500
gctgaagcag gccggcgacg tggaggagaa ccccggcccc atggccttac cagtgaccgc 1560
cttgctcctg ccgctggcct tgctgctcca cgccgccagg ccggatattc agatgaccca 1620
gagcccgagc agcctgagcg cgagcgtggg cgaccgcgtg accattacct gcaaagcgag 1680
ccaggatgtg ggcattgcgg tggcgtggta tcagcagaaa ccgggcaaag tgccgaaact 1740
gctgatttat tgggcgagca cccgccatac cggcgtgccg gatcgcttta gcggcagcgg 1800
cagcggcacc gattttaccc tgaccattag cagcctgcag ccggaagatg tggcgaccta 1860
ttattgccag cagtatagca gctatccgta tacctttggc cagggcacca aagtggaaat 1920
taaaggaggg gggggatccg ggggaggagg ctccggcgga ggcggaagcg aagtgcagct 1980
ggtggaaagc ggcggcggcc tggtgcagcc gggcggcagc ctgcgcctga gctgcgcggc 2040
gagcggcttt gattttagcc gctattggat gagctgggtg cgccaggcgc cgggcaaagg 2100
cctggaatgg attggcgaaa ttaacccgga tagcagcacc attaactatg cgccgagcct 2160
gaaagataaa tttattatta gccgcgataa cgcgaaaaac agcctgtatc tgcagatgaa 2220
cagcctgcgc gcggaagata ccgcggtgta ttattgcgcg cgcccggatg gcaactattg 2280
gtattttgat gtgtggggcc agggcaccct ggtgaccgtg agcagcacca cgacgccagc 2340
gccgcgacca ccaacaccgg cgcccaccat cgcgtcgcag cccctgtccc tgcgcccaga 2400
ggcgtgccgg ccagcggcgg ggggcgcagt gcacacgagg gggctggact tcgcctgtga 2460
tatctacatc tgggcgccct tggccgggac ttgtggggtc cttctcctgt cactggttat 2520
caccctttac tgcaaacggg gcagaaagaa actcctgtat atattcaagc aaccatttat 2580
gagaccagta caaactactc aagaggaaga tggctgtagc tgccgatttc cagaagaaga 2640
agaaggagga tgtgaactga gagtgaagtt cagcaggagc gcagacgccc ccgcgtacca 2700
gcagggccag aaccagctct ataacgagct caatctagga cgaagagagg agtacgatgt 2760
tttggacaag agacgtggcc gggaccctga gatgggggga aagccgcaga gaaggaagaa 2820
ccctcaggaa ggcctgtaca atgaactgca gaaagataag atggcggagg cctacagtga 2880
gattgggatg aaaggcgagc gccggagggg caaggggcac gatggccttt accagggtct 2940
cagtacagcc accaaggaca cctacgacgc ccttcacatg caggccctgc cccctcgcta 3000
agtttaaac 3009
<210> 23
<211> 423
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 23
agctagctgc agtaacgcca ttttgcaagg catggaaaaa taccaaacca agaatagaga 60
agttcagatc aagggcgggt acatgaaaat agctaacgtt gggccaaaca ggatatctgc 120
ggtgagcagt ttcggccccg gcccggggcc aagaacagat ggtcaccgca gtttcggccc 180
cggcccgagg ccaagaacag atggtcccca gatatggccc aaccctcagc agtttcttaa 240
gacccatcag atgtttccag gctcccccaa ggacctgaaa tgaccctgcg ccttatttga 300
attaaccaat cagcctgctt ctcgcttctg ttcgcgcgct tctgcttccc gagctctata 360
aaagagctca caacccctca ctcggcgcgc cagtcctccg acagactgag tcgcccgggt 420
acc 423
<210> 24
<211> 165
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 24
Met Ser Gly Leu Gly Arg Ser Arg Arg Gly Gly Arg Ser Arg Val Asp
1 5 10 15
Gln Glu Glu Arg Phe Pro Gln Gly Leu Trp Thr Gly Val Ala Met Arg
20 25 30
Ser Cys Pro Glu Glu Gln Tyr Trp Asp Pro Leu Leu Gly Thr Cys Met
35 40 45
Ser Cys Lys Thr Ile Cys Asn His Gln Ser Gln Arg Thr Cys Ala Ala
50 55 60
Phe Cys Arg Ser Leu Ser Cys Arg Lys Glu Gln Gly Lys Phe Tyr Asp
65 70 75 80
His Leu Leu Arg Asp Cys Ile Ser Cys Ala Ser Ile Cys Gly Gln His
85 90 95
Pro Lys Gln Cys Ala Tyr Phe Cys Glu Asn Lys Leu Arg Ser Pro Val
100 105 110
Asn Leu Pro Pro Glu Leu Arg Arg Gln Arg Ser Gly Glu Val Glu Asn
115 120 125
Asn Ser Asp Asn Ser Gly Arg Tyr Gln Gly Leu Glu His Arg Gly Ser
130 135 140
Glu Ala Ser Pro Ala Leu Pro Gly Leu Lys Leu Ser Ala Asp Gln Val
145 150 155 160
Ala Leu Val Tyr Ser
165
<210> 25
<211> 54
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 25
Met Leu Gln Met Ala Gly Gln Cys Ser Gln Asn Glu Tyr Phe Asp Ser
1 5 10 15
Leu Leu His Ala Cys Ile Pro Cys Gln Leu Arg Cys Ser Ser Asn Thr
20 25 30
Pro Pro Leu Thr Cys Gln Arg Tyr Cys Asn Ala Ser Val Thr Asn Ser
35 40 45
Val Lys Gly Thr Asn Ala
50
<210> 26
<211> 204
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 26
Ser Gly Pro Val Lys Glu Leu Val Gly Ser Val Gly Gly Ala Val Thr
1 5 10 15
Phe Pro Leu Lys Ser Lys Val Lys Gln Val Asp Ser Ile Val Trp Thr
20 25 30
Phe Asn Thr Thr Pro Leu Val Thr Ile Gln Pro Glu Gly Gly Thr Ile
35 40 45
Ile Val Thr Gln Asn Arg Asn Arg Glu Arg Val Asp Phe Pro Asp Gly
50 55 60
Gly Tyr Ser Leu Lys Leu Ser Lys Leu Lys Lys Asn Asp Ser Gly Ile
65 70 75 80
Tyr Tyr Val Gly Ile Tyr Ser Ser Ser Leu Gln Gln Pro Ser Thr Gln
85 90 95
Glu Tyr Val Leu His Val Tyr Glu His Leu Ser Lys Pro Lys Val Thr
100 105 110
Met Gly Leu Gln Ser Asn Lys Asn Gly Thr Cys Val Thr Asn Leu Thr
115 120 125
Cys Cys Met Glu His Gly Glu Glu Asp Val Ile Tyr Thr Trp Lys Ala
130 135 140
Leu Gly Gln Ala Ala Asn Glu Ser His Asn Gly Ser Ile Leu Pro Ile
145 150 155 160
Ser Trp Arg Trp Gly Glu Ser Asp Met Thr Phe Ile Cys Val Ala Arg
165 170 175
Asn Pro Val Ser Arg Asn Phe Ser Ser Pro Ile Leu Ala Arg Lys Leu
180 185 190
Cys Glu Gly Ala Ala Asp Asp Pro Asp Ser Ser Met
195 200
<210> 27
<211> 46
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 27
Met Asp Asp Ser Thr Glu Arg Glu Gln Ser Arg Leu Thr Ser Cys Leu
1 5 10 15
Lys Lys Arg Glu Glu Met Lys Leu Lys Glu Cys Val Ser Ile Leu Pro
20 25 30
Arg Lys Glu Ser Pro Ser Val Arg Ser Ser Lys Asp Gly Lys
35 40 45
<210> 28
<211> 242
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 28
Asp Pro Asn Phe Trp Leu Gln Val Gln Glu Ser Val Thr Val Gln Glu
1 5 10 15
Gly Leu Cys Val Leu Val Pro Cys Thr Phe Phe His Pro Ile Pro Tyr
20 25 30
Tyr Asp Lys Asn Ser Pro Val His Gly Tyr Trp Phe Arg Glu Gly Ala
35 40 45
Ile Ile Ser Arg Asp Ser Pro Val Ala Thr Asn Lys Leu Asp Gln Glu
50 55 60
Val Gln Glu Glu Thr Gln Gly Arg Phe Arg Leu Leu Gly Asp Pro Ser
65 70 75 80
Arg Asn Asn Cys Ser Leu Ser Ile Val Asp Ala Arg Arg Arg Asp Asn
85 90 95
Gly Ser Tyr Phe Phe Arg Met Glu Arg Gly Ser Thr Lys Tyr Ser Tyr
100 105 110
Lys Ser Pro Gln Leu Ser Val His Val Thr Asp Leu Thr His Arg Pro
115 120 125
Lys Ile Leu Ile Pro Gly Thr Leu Glu Pro Gly His Ser Lys Asn Leu
130 135 140
Thr Cys Ser Val Ser Trp Ala Cys Glu Gln Gly Thr Pro Pro Ile Phe
145 150 155 160
Ser Trp Leu Ser Ala Ala Pro Thr Ser Leu Gly Pro Arg Thr Thr His
165 170 175
Ser Ser Val Leu Ile Ile Thr Pro Arg Pro Gln Asp His Gly Thr Asn
180 185 190
Leu Thr Cys Gln Val Lys Phe Ala Gly Ala Gly Val Thr Thr Glu Arg
195 200 205
Thr Ile Gln Leu Asn Val Thr Tyr Val Pro Gln Asn Pro Thr Thr Gly
210 215 220
Ile Phe Pro Gly Asp Gly Ser Gly Lys Gln Glu Thr Arg Ala Gly Val
225 230 235 240
Val His
<210> 29
<211> 287
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 29
Thr Lys Glu Asp Pro Asn Pro Pro Ile Thr Asn Leu Arg Met Lys Ala
1 5 10 15
Lys Ala Gln Gln Leu Thr Trp Asp Leu Asn Arg Asn Val Thr Asp Ile
20 25 30
Glu Cys Val Lys Asp Ala Asp Tyr Ser Met Pro Ala Val Asn Asn Ser
35 40 45
Tyr Cys Gln Phe Gly Ala Ile Ser Leu Cys Glu Val Thr Asn Tyr Thr
50 55 60
Val Arg Val Ala Asn Pro Pro Phe Ser Thr Trp Ile Leu Phe Pro Glu
65 70 75 80
Asn Ser Gly Lys Pro Trp Ala Gly Ala Glu Asn Leu Thr Cys Trp Ile
85 90 95
His Asp Val Asp Phe Leu Ser Cys Ser Trp Ala Val Gly Pro Gly Ala
100 105 110
Pro Ala Asp Val Gln Tyr Asp Leu Tyr Leu Asn Val Ala Asn Arg Arg
115 120 125
Gln Gln Tyr Glu Cys Leu His Tyr Lys Thr Asp Ala Gln Gly Thr Arg
130 135 140
Ile Gly Cys Arg Phe Asp Asp Ile Ser Arg Leu Ser Ser Gly Ser Gln
145 150 155 160
Ser Ser His Ile Leu Val Arg Gly Arg Ser Ala Ala Phe Gly Ile Pro
165 170 175
Cys Thr Asp Lys Phe Val Val Phe Ser Gln Ile Glu Ile Leu Thr Pro
180 185 190
Pro Asn Met Thr Ala Lys Cys Asn Lys Thr His Ser Phe Met His Trp
195 200 205
Lys Met Arg Ser His Phe Asn Arg Lys Phe Arg Tyr Glu Leu Gln Ile
210 215 220
Gln Lys Arg Met Gln Pro Val Ile Thr Glu Gln Val Arg Asp Arg Thr
225 230 235 240
Ser Phe Gln Leu Leu Asn Pro Gly Thr Tyr Thr Val Gln Ile Arg Ala
245 250 255
Arg Glu Arg Val Tyr Glu Phe Leu Ser Ala Trp Ser Thr Pro Gln Arg
260 265 270
Phe Glu Cys Asp Gln Glu Glu Gly Ala Asn Thr Arg Ala Trp Arg
275 280 285
<210> 30
<211> 272
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 30
Pro Glu Glu Pro Leu Val Val Lys Val Glu Glu Gly Asp Asn Ala Val
1 5 10 15
Leu Gln Cys Leu Lys Gly Thr Ser Asp Gly Pro Thr Gln Gln Leu Thr
20 25 30
Trp Ser Arg Glu Ser Pro Leu Lys Pro Phe Leu Lys Leu Ser Leu Gly
35 40 45
Leu Pro Gly Leu Gly Ile His Met Arg Pro Leu Ala Ile Trp Leu Phe
50 55 60
Ile Phe Asn Val Ser Gln Gln Met Gly Gly Phe Tyr Leu Cys Gln Pro
65 70 75 80
Gly Pro Pro Ser Glu Lys Ala Trp Gln Pro Gly Trp Thr Val Asn Val
85 90 95
Glu Gly Ser Gly Glu Leu Phe Arg Trp Asn Val Ser Asp Leu Gly Gly
100 105 110
Leu Gly Cys Gly Leu Lys Asn Arg Ser Ser Glu Gly Pro Ser Ser Pro
115 120 125
Ser Gly Lys Leu Met Ser Pro Lys Leu Tyr Val Trp Ala Lys Asp Arg
130 135 140
Pro Glu Ile Trp Glu Gly Glu Pro Pro Cys Leu Pro Pro Arg Asp Ser
145 150 155 160
Leu Asn Gln Ser Leu Ser Gln Asp Leu Thr Met Ala Pro Gly Ser Thr
165 170 175
Leu Trp Leu Ser Cys Gly Val Pro Pro Asp Ser Val Ser Arg Gly Pro
180 185 190
Leu Ser Trp Thr His Val His Pro Lys Gly Pro Lys Ser Leu Leu Ser
195 200 205
Leu Glu Leu Lys Asp Asp Arg Pro Ala Arg Asp Met Trp Val Met Glu
210 215 220
Thr Gly Leu Leu Leu Pro Arg Ala Thr Ala Gln Asp Ala Gly Lys Tyr
225 230 235 240
Tyr Cys His Arg Gly Asn Leu Thr Met Ser Phe His Leu Glu Ile Thr
245 250 255
Ala Arg Pro Val Leu Trp His Trp Leu Leu Arg Thr Gly Gly Trp Lys
260 265 270
<210> 31
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 31
Pro Ile Cys Val Thr Val
1 5
<210> 32
<211> 47
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 32
Lys Ile Ser His Phe Leu Lys Met Glu Ser Leu Asn Phe Ile Arg Ala
1 5 10 15
His Thr Pro Tyr Ile Asn Ile Tyr Asn Cys Glu Pro Ala Asn Pro Ser
20 25 30
Glu Lys Asn Ser Pro Ser Thr Gln Tyr Cys Tyr Ser Ile Gln Ser
35 40 45
<210> 33
<211> 480
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 33
Asp Ser Ser Lys Trp Val Phe Glu His Pro Glu Thr Leu Tyr Ala Trp
1 5 10 15
Glu Gly Ala Cys Val Trp Ile Pro Cys Thr Tyr Arg Ala Leu Asp Gly
20 25 30
Asp Leu Glu Ser Phe Ile Leu Phe His Asn Pro Glu Tyr Asn Lys Asn
35 40 45
Thr Ser Lys Phe Asp Gly Thr Arg Leu Tyr Glu Ser Thr Lys Asp Gly
50 55 60
Lys Val Pro Ser Glu Gln Lys Arg Val Gln Phe Leu Gly Asp Lys Asn
65 70 75 80
Lys Asn Cys Thr Leu Ser Ile His Pro Val His Leu Asn Asp Ser Gly
85 90 95
Gln Leu Gly Leu Arg Met Glu Ser Lys Thr Glu Lys Trp Met Glu Arg
100 105 110
Ile His Leu Asn Val Ser Glu Arg Pro Phe Pro Pro His Ile Gln Leu
115 120 125
Pro Pro Glu Ile Gln Glu Ser Gln Glu Val Thr Leu Thr Cys Leu Leu
130 135 140
Asn Phe Ser Cys Tyr Gly Tyr Pro Ile Gln Leu Gln Trp Leu Leu Glu
145 150 155 160
Gly Val Pro Met Arg Gln Ala Ala Val Thr Ser Thr Ser Leu Thr Ile
165 170 175
Lys Ser Val Phe Thr Arg Ser Glu Leu Lys Phe Ser Pro Gln Trp Ser
180 185 190
His His Gly Lys Ile Val Thr Cys Gln Leu Gln Asp Ala Asp Gly Lys
195 200 205
Phe Leu Ser Asn Asp Thr Val Gln Leu Asn Val Lys His Thr Pro Lys
210 215 220
Leu Glu Ile Lys Val Thr Pro Ser Asp Ala Ile Val Arg Glu Gly Asp
225 230 235 240
Ser Val Thr Met Thr Cys Glu Val Ser Ser Ser Asn Pro Glu Tyr Thr
245 250 255
Thr Val Ser Trp Leu Lys Asp Gly Thr Ser Leu Lys Lys Gln Asn Thr
260 265 270
Phe Thr Leu Asn Leu Arg Glu Val Thr Lys Asp Gln Ser Gly Lys Tyr
275 280 285
Cys Cys Gln Val Ser Asn Asp Val Gly Pro Gly Arg Ser Glu Glu Val
290 295 300
Phe Leu Gln Val Gln Tyr Ala Pro Glu Pro Ser Thr Val Gln Ile Leu
305 310 315 320
His Ser Pro Ala Val Glu Gly Ser Gln Val Glu Phe Leu Cys Met Ser
325 330 335
Leu Ala Asn Pro Leu Pro Thr Asn Tyr Thr Trp Tyr His Asn Gly Lys
340 345 350
Glu Met Gln Gly Arg Thr Glu Glu Lys Val His Ile Pro Lys Ile Leu
355 360 365
Pro Trp His Ala Gly Thr Tyr Ser Cys Val Ala Glu Asn Ile Leu Gly
370 375 380
Thr Gly Gln Arg Gly Pro Gly Ala Glu Leu Asp Val Gln Tyr Pro Pro
385 390 395 400
Lys Lys Val Thr Thr Val Ile Gln Asn Pro Met Pro Ile Arg Glu Gly
405 410 415
Asp Thr Val Thr Leu Ser Cys Asn Tyr Asn Ser Ser Asn Pro Ser Val
420 425 430
Thr Arg Tyr Glu Trp Lys Pro His Gly Ala Trp Glu Glu Pro Ser Leu
435 440 445
Gly Val Leu Lys Ile Gln Asn Val Gly Trp Asp Asn Thr Thr Ile Ala
450 455 460
Cys Ala Ala Cys Asn Ser Trp Cys Ser Trp Ala Ser Pro Val Ala Leu
465 470 475 480
<210> 34
<211> 480
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 34
Gln Ser Pro Thr Pro Ser Pro Thr Gly Leu Thr Thr Ala Lys Met Pro
1 5 10 15
Ser Val Pro Leu Ser Ser Asp Pro Leu Pro Thr His Thr Thr Ala Phe
20 25 30
Ser Pro Ala Ser Thr Phe Glu Arg Glu Asn Asp Phe Ser Glu Thr Thr
35 40 45
Thr Ser Leu Ser Pro Asp Asn Thr Ser Thr Gln Val Ser Pro Asp Ser
50 55 60
Leu Asp Asn Ala Ser Ala Phe Asn Thr Thr Gly Val Ser Ser Val Gln
65 70 75 80
Thr Pro His Leu Pro Thr His Ala Asp Ser Gln Thr Pro Ser Ala Gly
85 90 95
Thr Asp Thr Gln Thr Phe Ser Gly Ser Ala Ala Asn Ala Lys Leu Asn
100 105 110
Pro Thr Pro Gly Ser Asn Ala Ile Ser Asp Val Pro Gly Glu Arg Ser
115 120 125
Thr Ala Ser Thr Phe Pro Thr Asp Pro Val Ser Pro Leu Thr Thr Thr
130 135 140
Leu Ser Leu Ala His His Ser Ser Ala Ala Leu Pro Ala Arg Thr Ser
145 150 155 160
Asn Thr Thr Ile Thr Ala Asn Thr Ser Asp Ala Tyr Leu Asn Ala Ser
165 170 175
Glu Thr Thr Thr Leu Ser Pro Ser Gly Ser Ala Val Ile Ser Thr Thr
180 185 190
Thr Ile Ala Thr Thr Pro Ser Lys Pro Thr Cys Asp Glu Lys Tyr Ala
195 200 205
Asn Ile Thr Val Asp Tyr Leu Tyr Asn Lys Glu Thr Lys Leu Phe Thr
210 215 220
Ala Lys Leu Asn Val Asn Glu Asn Val Glu Cys Gly Asn Asn Thr Cys
225 230 235 240
Thr Asn Asn Glu Val His Asn Leu Thr Glu Cys Lys Asn Ala Ser Val
245 250 255
Ser Ile Ser His Asn Ser Cys Thr Ala Pro Asp Lys Thr Leu Ile Leu
260 265 270
Asp Val Pro Pro Gly Val Glu Lys Phe Gln Leu His Asp Cys Thr Gln
275 280 285
Val Glu Lys Ala Asp Thr Thr Ile Cys Leu Lys Trp Lys Asn Ile Glu
290 295 300
Thr Phe Thr Cys Asp Thr Gln Asn Ile Thr Tyr Arg Phe Gln Cys Gly
305 310 315 320
Asn Met Ile Phe Asp Asn Lys Glu Ile Lys Leu Glu Asn Leu Glu Pro
325 330 335
Glu His Glu Tyr Lys Cys Asp Ser Glu Ile Leu Tyr Asn Asn His Lys
340 345 350
Phe Thr Asn Ala Ser Lys Ile Ile Lys Thr Asp Phe Gly Ser Pro Gly
355 360 365
Glu Pro Gln Ile Ile Phe Cys Arg Ser Glu Ala Ala His Gln Gly Val
370 375 380
Ile Thr Trp Asn Pro Pro Gln Arg Ser Phe His Asn Phe Thr Leu Cys
385 390 395 400
Tyr Ile Lys Glu Thr Glu Lys Asp Cys Leu Asn Leu Asp Lys Asn Leu
405 410 415
Ile Lys Tyr Asp Leu Gln Asn Leu Lys Pro Tyr Thr Lys Tyr Val Leu
420 425 430
Ser Leu His Ala Tyr Ile Ile Ala Lys Val Gln Arg Asn Gly Ser Ala
435 440 445
Ala Met Cys His Phe Thr Thr Lys Ser Ala Pro Pro Ser Gln Val Trp
450 455 460
Asn Met Thr Val Ser Met Thr Ser Asp Asn Ser Met His Val Lys Cys
465 470 475 480
<210> 35
<211> 65
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 35
Ile Thr Cys Pro Pro Pro Met Ser Val Glu His Ala Asp Ile Trp Val
1 5 10 15
Lys Ser Tyr Ser Leu Tyr Ser Arg Glu Arg Tyr Ile Cys Asn Ser Gly
20 25 30
Phe Lys Arg Lys Ala Gly Thr Ser Ser Leu Thr Glu Cys Val Leu Asn
35 40 45
Lys Ala Thr Asn Val Ala His Trp Thr Thr Pro Ser Leu Lys Cys Ile
50 55 60
Arg
65
<210> 36
<211> 153
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic sequence
<400> 36
Met Tyr Arg Met Gln Leu Leu Ser Cys Ile Ala Leu Ser Leu Ala Leu
1 5 10 15
Val Thr Asn Ser Gly Ile His Val Phe Ile Leu Gly Cys Phe Ser Ala
20 25 30
Gly Leu Pro Lys Thr Glu Ala Asn Trp Val Asn Val Ile Ser Asp Leu
35 40 45
Lys Lys Ile Glu Asp Leu Ile Gln Ser Met His Ile Asp Ala Thr Leu
50 55 60
Tyr Thr Glu Ser Asp Val His Pro Ser Cys Lys Val Thr Ala Met Lys
65 70 75 80
Cys Phe Leu Leu Glu Leu Gln Val Ile Ser Leu Glu Ser Gly Asp Ala
85 90 95
Ser Ile His Asp Thr Val Glu Asn Leu Ile Ile Leu Ala Asn Asn Ser
100 105 110
Leu Ser Ser Asn Gly Asn Val Thr Glu Ser Gly Cys Lys Glu Cys Glu
115 120 125
Glu Leu Glu Glu Lys Asn Ile Lys Glu Phe Leu Gln Ser Phe Val His
130 135 140
Ile Val Gln Met Phe Ile Asn Thr Ser
145 150
Claims (75)
- (i.) 제1 항원 인식 도메인, 제1 신호 펩타이드, 제1 힌지 영역, 제1 막통과(transmembrane) 도메인, 제1 보조 자극 도메인, 그리고 제1 신호전달 도메인을 포함하는 제1 키메라 항원 수용체 폴리펩타이드; 및
(ii.) 제2 항원 인식 도메인, 제2 신호 펩타이드, 제2 힌지 영역, 제2 막통과 도메인, 제2 보조 자극 도메인, 그리고 제2 신호전달 도메인을 포함하는 제2 키메라 항원 수용체 폴리펩타이드
를 포함하며, 여기서 제1 항원 인식 도메인 및 제2 항원 인식 도메인은 상이한 것인, 조작된 세포. - 제1항에 있어서, 제1 키메라 항원 수용체 폴리펩타이드 및 제2 키메라 조작된 폴리펩타이드는 단일 폴리펩타이드 분자 상에 존재하고, 고효율 절단 부위를 포함하는 아미노산 서열은 제1 키메라 항원 수용체 폴리펩타이드와 제2 키메라 항원 수용체 폴리펩타이드 사이에 배치되는 것인, 조작된 세포.
- 제2항에 있어서, 고효율 절단 부위는 P2A, T2A, E2A, 및 F2A로 이루어진 그룹으로부터 선택되는 것인, 조작된 세포.
- 제1항 내지 제3항 중 어느 한 항에 있어서, 제1 보조-자극 도메인 및 제2 보조-자극 도메인은 상이한 것인, 조작된 세포.
- 제1항 내지 제4항 중 어느 한 항에 있어서, 제1 보조 자극 도메인은 CD28을 포함하고, 제2 보조 자극 도메인은 4-1BB를 포함하는 것인, 조작된 세포.
- 제1항 내지 제5항 중 어느 한 항에 있어서, 제1 항원 인식 도메인의 표적은 인터루킨 6 수용체, NY-ESO-1, 알파 태아단백 (AFP), 글리피칸-3 (GPC3), BCMA, BAFF-R, BCMA, TACI, LeY, CD5, CD13, CD14, CD15, CD19, CD20, CD22, CD33, CD41, CD61, CD64, CD68, CD117, CD123, CD138, CD267, CD269, CD38, Flt3 수용체, 및 CS1로 이루어진 그룹으로부터 선택되고, 제2 항원 인식 도메인의 표적은 인터루킨 6 수용체, NY-ESO-1, 알파 태아단백 (AFP), 글리피칸-3 (GPC3), BCMA, BAFF-R, BCMA, TACI, LeY, CD5, CD13, CD14, CD15, CD19, CD20, CD22, CD33, CD41, CD61, CD64, CD68, CD117, CD123, CD138, CD267, CD269, CD38, Flt3 수용체, 및 CS1로 이루어진 그룹으로부터 선택되는 것인, 조작된 세포.
- 제1항 내지 제6항 중 어느 한 항에 있어서, 제1 항원 인식 도메인의 표적은 CD267 또는 CD269를 포함하고, 제2 항원 인식 도메인의 표적은 CD19, CD38, CD138, CD138 및 CS1로 이루어진 그룹으로부터 선택되는 것인, 조작된 세포.
- 제1항 내지 제6항 중 어느 한 항에 있어서, 제1 항원 인식 도메인의 표적은 CD19를 포함하고, 제2 항원 인식 도메인의 표적은 CD20, CD22, CD33, CD123, CD267, CD269, CD38 및 CS1로 이루어진 그룹으로부터 선택되는 것인, 조작된 세포.
- 제1항 내지 제6항 중 어느 한 항에 있어서, 제1 항원 인식 도메인의 표적은 CD19를 포함하고, 제2 항원 인식 도메인의 표적은 CD20, CD22 및 CD123으로 이루어진 그룹으로부터 선택되는 것인, 조작된 세포.
- 제1항 내지 제6항 중 어느 한 항에 있어서, 제1 항원 인식 도메인의 표적은 CD33을 포함하고, 제2 항원 인식 도메인의 표적은 LeY 또는 CD123을 포함하는 것인, 조작된 세포.
- 제1항 내지 제6항 중 어느 한 항에 있어서, 제1 항원 인식 도메인의 표적은 BCMA를 포함하고, 제2 항원 인식 도메인의 표적은 CS1, CD19, CD38, CD138 또는 CS1을 포함하는 것인, 조작된 세포.
- 제1항 내지 제11항 중 어느 한 항에 있어서, T 세포 또는 자연 살상(Natural Killer) 세포인, 조작된 세포.
- 제12항에 있어서, T 세포는 CD4 T 세포 또는 CD8 T 세포인, 조작된 세포.
- 제12항에 있어서, 자연 살상 세포는 NKT 세포 또는 NK-92 세포인, 조작된 세포.
- 키메라 항원 수용체 폴리펩타이드 및 인핸서를 포함하는 조작된 폴리펩타이드.
- 제15항에 있어서, 키메라 항원 수용체 폴리펩타이드는 CD2, CD3, CD4, CD5, CD7, CD8, CD45 및 CD52로 이루어진 그룹으로부터 선택된 표적에 대해 선택적인 하나의 항원 인식 도메인을 포함하는 것인, 조작된 폴리펩타이드.
- 제15항에 있어서, 키메라 항원 수용체 폴리펩타이드는 CD19, CD20, CD22, CD33, CD38, CD123, CD138, CD267, CD269, CD38 및 CS1로 이루어진 그룹으로부터 선택된 표적에 대해 선택적인 하나의 항원 인식 도메인을 포함하는 것인, 조작된 폴리펩타이드.
- 제15항에 있어서, 키메라 항원 수용체 폴리펩타이드는 CD45 항원 인식 도메인을 포함하는 것인, 조작된 폴리펩타이드.
- 제15항 내지 제18항 중 어느 한 항에 있어서, 상기 인핸서는 PD-1, PD-L1, CSF1R, CTAL-4, TIM-3, TGFR 베타, IL-2, IL-7, IL-12, IL-15, IL-21, 이들의 기능적 단편, 및 이들의 조합물로 이루어진 그룹으로부터 선택되는 것인, 조작된 폴리펩타이드.
- 제15항 내지 제18항 중 어느 한 항에 있어서, 인핸서 수용체 또는 이의 기능적 단편을 추가로 포함하는 조작된 폴리펩타이드.
- 제20항에 있어서, 인핸서 수용체는 IL-15RA 또는 이의 기능적 단편을 포함하는 조작된 폴리펩타이드.
- 제21항에 있어서, 기능적 단편은 스시 (sushi) 도메인을 포함하는 것인, 조작된 폴리펩타이드.
- 제15항 내지 제22항 중 어느 한 항에 있어서, 키메라 항원 수용체 폴리펩타이드 및 인핸서는 단일 폴리펩타이드 분자 상에 존재하는 것인, 조작된 폴리펩타이드.
- 제23항에 있어서, 고효율 절단 부위는 키메라 항원 수용체와 인핸서 사이에 배치되는 것인, 조작된 폴리펩타이드.
- 제24항에 있어서, 고효율 절단 부위는 P2A, T2A, E2A 및 F2A로 이루어진 그룹으로부터 선택되는 것인, 조작된 폴리펩타이드.
- 제15항 내지 제25항 중 어느 한 항에 있어서, 키메라 항원 수용체 폴리펩타이드는 CD2, CD4 및 CD19로 이루어진 그룹으로부터 선택된 표적에 대해 선택적인 하나의 항원 인식 도메인을 포함하고, 인핸서는 IL-15 또는 IL-15RA를 포함하는 것인, 조작된 폴리펩타이드.
- 제15항 내지 제26항 중 어느 한 항에 따른 폴리펩타이드를 코딩하는 조작된 폴리뉴클레오타이드.
- 제27항에 있어서, 벡터 안에 존재하는 조작된 폴리뉴클레오타이드.
- 제15항 내지 제26항 중 어느 한 항에 따른 폴리펩타이드를 포함하는 조작된 세포.
- 제24항 또는 제25항에 따른 폴리뉴클레오타이드를 포함하는 조작된 세포.
- 제29항 또는 제30항에 있어서, T 세포 또는 자연 살상 세포를 포함하는 조작된 세포.
- 제31항에 있어서, T 세포 또는 자연 살상 세포인 조작된 세포.
- 제32항에 있어서, T 세포는 CD4 또는 CD8 T 세포인, 조작된 세포.
- 제31항에 있어서, 자연 살상 세포는 NKT 세포 또는 NK-92 세포인, 조작된 세포.
- 제1항 내지 제15항 및 제29항 내지 제34항 중 어느 한 항에 따른 조작된 세포를 B 세포 림프종의 치료를 필요로 하는 환자에게 투여하는 것을 포함하는 B 세포 림프종의 치료 방법.
- 제1항 내지 제15항 및 제29항 내지 제34항 중 어느 한 항에 따른 조작된 세포를 T 세포 림프종의 치료를 필요로 하는 환자에게 투여하는 것을 포함하는 T 세포 림프종의 치료 방법.
- 제1항 내지 제15항 및 제29항 내지 제34항 중 어느 한 항에 따른 조작된 세포를 다발성 골수종의 치료를 필요로 하는 환자에게 투여하는 것을 포함하는 다발성 골수종의 치료 방법.
- 제1항 내지 제15항 및 제29항 내지 제34항 중 어느 한 항에 따른 조작된 세포를 만성 골수성 백혈병의 치료를 필요로 하는 환자에게 투여하는 것을 포함하는 만성 골수성 백혈병의 치료 방법이며, 여기서 제1 항원 인식 도메인의 표적은 CD33을 포함하고, 제2 항원 인식 도메인의 표적은 CD123을 포함하는 것인, 만성 골수성 백혈병의 치료 방법.
- 제1항 내지 제15항 중 어느 한 항에 따른 조작된 세포를 B-세포 급성 림프모구성 백혈병 (B-ALL)의 치료를 필요로 하는 환자에게 투여하는 것을 포함하는 B-세포 급성 림프모구성 백혈병 (B-ALL)의 치료 방법이며, 여기서 제1 항원 인식 도메인의 표적은 CD19를 포함하고, 제2 항원 인식 도메인의 표적은 CD123을 포함하는 것인, B-세포 급성 림프모구성 백혈병 (B-ALL)의 치료 방법.
- 제1항 내지 제6항 중 어느 한 항에 따른 조작된 세포를 다발성 골수종의 치료를 필요로 하는 환자에게 투여하는 것을 포함하는 다발성 골수종의 치료 방법이며, 여기서 제1 항원 인식 도메인의 표적은 CD38, CS1, BCMA, 및 CD38로 이루어진 그룹으로부터 선택되고, 제2 항원 인식 도메인의 표적은 CD38, CS1, BCMA, 및 CD38로 이루어진 그룹으로부터 선택되는 것인, 다발성 골수종의 치료 방법.
- 제1항 내지 제15항 중 어느 한 항에 따른 조작된 세포를 세포 증식성 질환의 치료를 필요로 하는 환자에게 투여하는 것을 포함하는 세포 증식성 질환의 치료 방법이며, 여기서 제1 항원 인식 도메인의 표적은 BCMA, TAC1, CS1, 및 BAFF-R로 이루어진 그룹으로부터 선택되고, 제2 항원 인식 도메인의 표적은 BCMA, TAC1, CS1, 및 BAFF-R로 이루어진 그룹으로부터 선택되는 것인, 세포 증식성 질환의 치료 방법.
- 제41항에 있어서, 세포 증식성 질환은 림프종, 백혈병 및 형질세포종양로 이루어진 그룹으로부터 선택되는 것인, 세포 증식성 질환의 치료 방법.
- 제42항에 있어서, 형질세포종양은 형질세포성 백혈병, 다발성 골수종, 형질세포종, 아밀로이드증, 발덴스트롬 마크로글로불린혈증, 중쇄 질환, 고립성 골 형질세포종, 미확정 단클론 감마글로불린혈증 (MGUS), 그리고 무증상다발성골수종으로부터 선택되는 것인, 세포 증식성 질환의 치료 방법.
- 제30항 내지 제34항 중 어느 한 항에 따른 조작된 세포를 세포 증식성 질환의 치료를 필요로 하는 환자에게 투여하는 것을 포함하는 세포증식성 질환의 치료 방법.
- 인터루킨 6 수용체, NY-ESO-1, 알파 태아단백 (AFP), 글리피칸-3 (GPC3), BCMA, BAFF-R, TACI, LeY, CD5, CD13, CD14, CD15, CD45, CD19, CD20, CD22, CD33, CD41, CD61, CD64, CD68, CD117, CD123, CD138, CD267, CD269, CD38, Flt3 수용체, 및 CS1로 이루어진 그룹으로부터 선택된 표적에 대해 선택적인 항원 인식 도메인을 갖는 키메라 항원 수용체 폴리펩타이드를 포함하는 조작된 세포 및 CAR 증폭제를 세포증식성 질환의 치료를 필요로 하는 환자에게 투여하는 것을 포함하는 세포증식성 질환의 치료 방법.
- 제45항에 있어서, 상기 CAR 증폭제는 면역 체크 포인트 경로, 콜로니자극인자-1 수용체 (CSF1R)의 억제제, PD-1, PD-L1, IL-2, IL-12, IL-15, CSF1R, CTAL-4, TIM-3, 및 TGFR 베타를 표적으로 하는 제제로 이루어진 그룹으로부터 선택되는 것인, 세포증식성 질환의 치료 방법.
- 제13항 내지 제23항 중 어느 한 항에 따른 조작된 폴리펩타이드를 포함하는 조작된 세포를 세포증식성 질환의 치료를 필요로 하는 환자에게 투여하는 것을 포함하는 세포증식성 질환의 치료 방법.
- 신호 펩타이드, CD45 항원 인식 도메인, 힌지 영역, 막통과 도메인, 적어도 하나의 보조 자극 도메인, 및 신호전달 도메인을 포함하는 조작된 키메라 항원 수용체 폴리펩타이드.
- 제48항에 있어서, 상기 CD45 항원 인식 도메인은 CD45에 대해 선택적인 단클론항체의 결합 부분 또는 가변 영역을 포함하는 것인, 조작된 키메라 항원 수용체 폴리펩타이드.
- 제48항 또는 제49항에 있어서, 상기 CD45 항원 인식 도메인은 CD45 scFv를 포함하는 것인, 조작된 키메라 항원 수용체 폴리펩타이드.
- 제48항 내지 제50항 중 어느 한 항에 있어서, 상기 힌지 영역은 CD-8 알파, CD28, 4-1BB, OX40, CD3-제타, 이들의 기능성 유도체, 및 이들의 조합물로 이루어진 그룹으로부터 선택된 인간 단백질의 힌지 영역을 포함하는 것인, 조작된 키메라 항원 수용체 폴리펩타이드.
- 제48항 내지 제51항 중 어느 한 항에 있어서, 상기 막통과 도메인은 CD-8 알파, CD28, 4-1BB, OX40, CD3-제타, 이들의 기능성 유도체, 및 이들의 조합물로 이루어진 그룹으로부터 선택된 인간 단백질의 막통과 영역을 포함하는 것인, 조작된 키메라 항원 수용체 폴리펩타이드.
- 제48항 내지 제52항 중 어느 한 항에 있어서, 상기 신호전달 도메인은 CD3 제타, 공통 FcR 감마 (FCER1G), Fc 감마 Rlla, FcR 베타 (Fc 엡실론 립), CD3 감마, CD3 델타, CD3 엡실론, CD79a, CD79b, DAP10, DAP12, 이들의 활성 단편 및 이들의 조합물로 이루어진 그룹으로부터 선택된 신호전달 도메인을 포함하는 것인, 조작된 키메라 항원 수용체 폴리펩타이드.
- 제48항 내지 제53항 중 어느 한 항에 있어서, 상기 보조 자극 도메인은 OX40, CD27, CD28, CD30, CD40, PD-1, CD2, CD7, CD258, NKG2C, NKG2D, B7-H3; CD83, ICAM-1, LFA-1 (CD1 1a/CD18), ICOS, 및 4-1BB (CD137)에 결합하는 리간드; 이들의 활성 단편; 및 이들의 조합물로 이루어진 그룹으로부터 선택된 단백질로부터의 보조 자극 도메인을 포함하는 것인, 조작된 키메라 항원 수용체 폴리펩타이드.
- 제48항 내지 제54항 중 어느 한 항에 따른 폴리펩타이드를 코딩하는 조작된 폴리뉴클레오타이드.
- 제55항에 있어서, 벡터 안에 존재하는 조작된 폴리뉴클레오타이드.
- 제55항 또는 제56항에 따른 폴리뉴클레오타이드를 포함하는 조작된 세포.
- 제48항 내지 제54항 중 어느 한 항에 따른 폴리펩타이드를 포함하는 조작된 세포.
- 제57항 또는 제58항에 있어서, T 세포 또는 자연 살상 세포인 조작된 세포.
- 제59항에 있어서, T 세포는 CD4 T 세포 또는 CD8 T 세포인, 조작된 세포.
- 제59항에 있어서, 자연 살상 세포는 NKT 세포 또는 NK-92 세포인, 조작된 세포.
- i. 표적 세포를 제1항 내지 제14항, 제29항 내지 제34항, 및 제57항 내지 제61항 중 어느 한 항에 따른 조작된 세포의 유효량과 접촉시키는 단계; 및
ii. 선택적으로, 상기 세포 수의 감소를 검정하는 단계
를 포함하는, 표적 세포의 수를 줄이는 방법이며, 여기서 상기 표적 세포는 인터루킨 6 수용체, NY-ESO-1, 알파 태아단백 (AFP), 글리피칸-3 (GPC3), BCMA, BAFF-R, TACI, LeY, CD5, CD13, CD14, CD15, CD19, CD20, CD22, CD33, CD41, CD45, CD61, CD64, CD68, CD117, CD123, CD138, CD267, CD269, CD38, Flt3 수용체, 및 CS1로 이루어진 그룹으로부터 선택된 적어도 하나의 세포 표면 항원을 포함하는 것인, 표적 세포의 수를 줄이는 방법. - 제1항 내지 제6항 중 어느 한 항에 있어서, 제1 항원 인식 도메인의 표적은 BCMA를 포함하고, 제2 항원 인식 도메인은 CS1을 포함하는 것인, 조작된 세포.
- 제1항 내지 제6항 중 어느 한 항에 있어서, 제1 항원 인식 도메인의 표적은 CD19를 포함하고, 제2 항원 인식 도메인은 BCMA를 포함하는 것인, 조작된 세포.
- 제1항 내지 제6항 중 어느 한 항에 있어서, 제1 항원 인식 도메인의 표적은 CD19를 포함하고, 제2 항원 인식 도메인의 표적은 CD22를 포함하는 것인, 조작된 세포.
- 제1항 내지 제6항 중 어느 한 항에 있어서, 제1 항원 인식 도메인의 표적은 CD19를 포함하고, 제2 항원 인식 도메인은 CD20을 포함하는 것인, 조작된 세포.
- 제1항 내지 제6항 중 어느 한 항에 있어서, 제1 항원 인식 도메인의 표적은 CD19를 포함하고, 제2 항원 인식 도메인은 CD123을 포함하는 것인, 조작된 세포.
- 제1항 내지 제6항 중 어느 한 항에 있어서, 제1 항원 인식 도메인의 표적은 CD33을 포함하고, 제2 항원 인식 도메인의 표적은 CD123을 포함하는 것인, 조작된 세포.
- 제1항 내지 제6항 중 어느 한 항에 있어서, 제1 항원 인식 도메인의 표적은 CD269를 포함하고, 제2 항원 인식 도메인의 표적은 CS1을 포함하는 것인, 조작된 세포.
- 제29항 내지 제34항 중 어느 한 항에 있어서, 항원 인식 도메인의 표적은 CD4를 포함하고, 인핸서는 IL-15RA를 포함하는 것인, 조작된 세포.
- 제1항 내지 제6항 중 어느 한 항에 있어서, TAC1 항원 인식 도메인은 APRIL 리간드 또는 BAFF 리간드 또는 이의 일부를 포함하는 것인, 조작된 세포.
- 제1항 내지 제6항 중 어느 한 항에 있어서, BCMA 항원 인식 도메인은 APRIL 리간드 또는 BAFF 리간드 또는 이의 일부를 포함하는 것인, 조작된 세포.
- 제1항 내지 제6항 중 어느 한 항에 있어서, BAFF-R 항원 인식 도메인은 BAFF 리간드 또는 이의 일부를 포함하는 것인, 조작된 세포.
- 제1항 내지 제6항 중 어느 한 항에 있어서, 제1 보조 자극 도메인 및 제2 보조 자극 도메인은 동일한 것인, 조작된 세포.
- 제1항 내지 제6항 및 제11항 중 어느 한 항에 있어서, 제1 보조 자극 도메인 및 제2 보조 자극 도메인은 4-1BB 보조 자극 도메인을 포함하는 것인, 조작된 세포.
Applications Claiming Priority (7)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201562184321P | 2015-06-25 | 2015-06-25 | |
| US62/184,321 | 2015-06-25 | ||
| US201562235840P | 2015-10-01 | 2015-10-01 | |
| US62/235,840 | 2015-10-01 | ||
| US201562244435P | 2015-10-21 | 2015-10-21 | |
| US62/244,435 | 2015-10-21 | ||
| PCT/US2016/039306 WO2016210293A1 (en) | 2015-06-25 | 2016-06-24 | CHIMERIC ANTIGEN RECEPTORS (CARs), COMPOSITIONS AND METHODS OF USE THEREOF |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20180021137A true KR20180021137A (ko) | 2018-02-28 |
Family
ID=57586695
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020187002389A KR20180021137A (ko) | 2015-06-25 | 2016-06-24 | 키메라 항원 수용체 (car), 조성물 및 이의 사용 방법 |
Country Status (10)
| Country | Link |
|---|---|
| US (1) | US11655452B2 (ko) |
| EP (1) | EP3313872A4 (ko) |
| JP (3) | JP6961497B2 (ko) |
| KR (1) | KR20180021137A (ko) |
| CN (2) | CN107849112B (ko) |
| AU (3) | AU2016283102B2 (ko) |
| CA (1) | CA2990177A1 (ko) |
| IL (2) | IL256410B (ko) |
| SG (1) | SG10201913682QA (ko) |
| WO (1) | WO2016210293A1 (ko) |
Cited By (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2021225399A1 (ko) * | 2020-05-08 | 2021-11-11 | (주)에스엠티바이오 | 암 치료를 위한 키메라 항원 수용체 |
Families Citing this family (125)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| SG11201505858VA (en) | 2013-01-28 | 2015-09-29 | St Jude Childrens Res Hospital | A chimeric receptor with nkg2d specificity for use in cell therapy against cancer and infectious disease |
| CN112175911B (zh) | 2014-05-15 | 2023-10-13 | 新加坡国立大学 | 经修饰的自然杀伤细胞及其用途 |
| CN107002045A (zh) | 2014-12-24 | 2017-08-01 | Ucl商务股份有限公司 | 细胞 |
| US11173179B2 (en) | 2015-06-25 | 2021-11-16 | Icell Gene Therapeutics Llc | Chimeric antigen receptor (CAR) targeting multiple antigens, compositions and methods of use thereof |
| JP6961497B2 (ja) | 2015-06-25 | 2021-11-05 | アイセル・ジーン・セラピューティクス・エルエルシー | キメラ抗体受容体(CARs)の構成およびその使用方法 |
| WO2017180587A2 (en) | 2016-04-11 | 2017-10-19 | Obsidian Therapeutics, Inc. | Regulated biocircuit systems |
| US11293021B1 (en) | 2016-06-23 | 2022-04-05 | Inscripta, Inc. | Automated cell processing methods, modules, instruments, and systems |
| US10253316B2 (en) | 2017-06-30 | 2019-04-09 | Inscripta, Inc. | Automated cell processing methods, modules, instruments, and systems |
| EP3474867A4 (en) | 2016-06-24 | 2020-05-20 | iCell Gene Therapeutics LLC | CHIMERIC ANTIGEN (CAR) RECEPTORS, COMPOSITIONS AND RELATED METHODS |
| US11078481B1 (en) * | 2016-08-03 | 2021-08-03 | KSQ Therapeutics, Inc. | Methods for screening for cancer targets |
| EP4282969A3 (en) * | 2016-09-02 | 2024-01-31 | Lentigen Technology, Inc. | Compositions and methods for treating cancer with duocars |
| CN110114371A (zh) * | 2016-11-11 | 2019-08-09 | 奥托路斯有限公司 | 嵌合抗原受体 |
| JP7291396B2 (ja) * | 2016-11-22 | 2023-06-15 | ティーシーアール2 セラピューティクス インク. | 融合タンパク質を用いたtcrの再プログラミングのための組成物及び方法 |
| KR20190114966A (ko) | 2016-12-09 | 2019-10-10 | 온키뮨 리미티드 | 조작된 자연 살해 세포 및 이의 용도 |
| WO2018128485A1 (ko) | 2017-01-05 | 2018-07-12 | 한국생명공학연구원 | 항-코티닌 키메릭 항원 수용체를 발현하는 자연살해 세포 |
| US20190375815A1 (en) | 2017-01-31 | 2019-12-12 | Novartis Ag | Treatment of cancer using chimeric t cell receptor proteins having multiple specificities |
| WO2018144777A2 (en) * | 2017-02-01 | 2018-08-09 | Nant Holdings Ip, Llc | Calreticulin-mediated cancer treatment |
| CN110650743B (zh) | 2017-03-13 | 2023-12-29 | 波赛达治疗公司 | 用于选择性消除和替换造血干细胞的组合物和方法 |
| US11896616B2 (en) | 2017-03-27 | 2024-02-13 | National University Of Singapore | Stimulatory cell lines for ex vivo expansion and activation of natural killer cells |
| CA3056439A1 (en) | 2017-03-27 | 2018-10-04 | National University Of Singapore | Truncated nkg2d chimeric receptors and uses thereof in natural killer cell immunotherapy |
| EP3612568B8 (en) * | 2017-04-18 | 2021-12-08 | Autolus Limited | Cell |
| WO2018201056A1 (en) | 2017-04-28 | 2018-11-01 | Novartis Ag | Cells expressing a bcma-targeting chimeric antigen receptor, and combination therapy with a gamma secretase inhibitor |
| US20200179511A1 (en) | 2017-04-28 | 2020-06-11 | Novartis Ag | Bcma-targeting agent, and combination therapy with a gamma secretase inhibitor |
| WO2018213337A1 (en) * | 2017-05-15 | 2018-11-22 | The United States Of America, As Represented By The Secretary, Department Of Health And Human Services | Bicistronic chimeric antigen receptors and their uses |
| CA3063169A1 (en) * | 2017-06-07 | 2018-12-13 | The General Hospital Corporation | T cells expressing a chimeric antigen receptor |
| CN111107866A (zh) | 2017-06-12 | 2020-05-05 | 黑曜石疗法公司 | 用于免疫疗法的pde5组合物和方法 |
| US20200223918A1 (en) * | 2017-06-21 | 2020-07-16 | Icell Gene Therapeutics Llc | CHIMERIC ANTIGEN RECEPTORS (CARs), COMPOSITIONS AND METHODS THEREOF |
| US9982279B1 (en) | 2017-06-23 | 2018-05-29 | Inscripta, Inc. | Nucleic acid-guided nucleases |
| US10011849B1 (en) | 2017-06-23 | 2018-07-03 | Inscripta, Inc. | Nucleic acid-guided nucleases |
| CN111315872A (zh) * | 2017-06-30 | 2020-06-19 | 纪念斯隆-凯特林癌症中心 | 用于癌症的过继性细胞疗法的组合物和方法 |
| US20200306301A1 (en) | 2017-07-03 | 2020-10-01 | Torque Therapeutics, Inc. | Polynucleotides Encoding Immunostimulatory Fusion Molecules and Uses Thereof |
| US20200165348A1 (en) * | 2017-07-20 | 2020-05-28 | H. Lee Moffitt Cancer Center And Research Institute, Inc. | Cd123-binding chimeric antigen receptors |
| JP2021520184A (ja) * | 2017-09-29 | 2021-08-19 | ナントセル,インコーポレイテッド | Cd1d及びtcr−nkt細胞 |
| EP3694886A4 (en) * | 2017-10-12 | 2021-09-22 | iCell Gene Therapeutics, LLC | COMPOSITE CHIMERIC ANTIGEN RECEPTOR (CCAR) TARGETING MULTIPLE ANTIGENS, COMPOSITIONS AND METHOD OF USING THEREOF |
| JP2021501570A (ja) | 2017-10-18 | 2021-01-21 | ノバルティス アーゲー | 選択的タンパク質分解のための組成物及び方法 |
| US20210187024A1 (en) * | 2017-11-01 | 2021-06-24 | Nantkwest, Inc. | NK-92 Cells to Stimulate Anti-Cancer Vaccine |
| WO2019099639A1 (en) | 2017-11-15 | 2019-05-23 | Navartis Ag | Bcma-targeting chimeric antigen receptor, cd19-targeting chimeric antigen receptor, and combination therapies |
| MX2020005651A (es) | 2017-11-30 | 2020-10-28 | Novartis Ag | Receptor de antigeno quimerico dirigido a bcma y usos del mismo. |
| CN111587254A (zh) * | 2017-12-05 | 2020-08-25 | 特拉维夫医疗中心医学研究基础设施及卫生服务基金 | 包含抗cd38和抗cd138嵌合抗原受体的t-细胞及其用途 |
| EP3737408A1 (en) | 2018-01-08 | 2020-11-18 | Novartis AG | Immune-enhancing rnas for combination with chimeric antigen receptor therapy |
| WO2019152660A1 (en) | 2018-01-31 | 2019-08-08 | Novartis Ag | Combination therapy using a chimeric antigen receptor |
| EP3752203A1 (en) | 2018-02-13 | 2020-12-23 | Novartis AG | Chimeric antigen receptor therapy in combination with il-15r and il15 |
| SG11202007755YA (en) * | 2018-02-23 | 2020-09-29 | H Lee Moffitt Cancer Ct & Res | Cd83-binding chimeric antigen receptors |
| CA3093078A1 (en) | 2018-03-06 | 2019-09-12 | The Trustees Of The University Of Pennsylvania | Prostate-specific membrane antigen cars and methods of use thereof |
| CN111954527A (zh) * | 2018-04-09 | 2020-11-17 | 梅约医学教育与研究基金会 | 治疗移植物抗宿主病的方法和材料 |
| US10869888B2 (en) | 2018-04-17 | 2020-12-22 | Innovative Cellular Therapeutics CO., LTD. | Modified cell expansion and uses thereof |
| US10526598B2 (en) | 2018-04-24 | 2020-01-07 | Inscripta, Inc. | Methods for identifying T-cell receptor antigens |
| US10858761B2 (en) | 2018-04-24 | 2020-12-08 | Inscripta, Inc. | Nucleic acid-guided editing of exogenous polynucleotides in heterologous cells |
| EP3561053A1 (en) * | 2018-04-26 | 2019-10-30 | Baylor College of Medicine | Immune effector cells and molecular adaptors with an antigen cytokine complex for effective cancer immunotherapy |
| EP3784272A1 (en) * | 2018-04-27 | 2021-03-03 | CRISPR Therapeutics AG | Anti-bcma car-t-cells for plasma cell depletion |
| EA202092945A1 (ru) * | 2018-06-01 | 2021-04-29 | Юниверсити Оф Саутерн Калифорния | Различные антигенсвязывающие домены, новые платформы и другие улучшения для клеточной терапии |
| CN110577605A (zh) * | 2018-06-11 | 2019-12-17 | 浙江启新生物技术有限公司 | 一种靶向多发性骨髓瘤多种抗原的嵌合抗原受体t(car-t)细胞的构建方法及其应用 |
| CN110305847B (zh) * | 2018-07-27 | 2021-05-18 | 赛诺生(深圳)基因产业发展有限公司 | 一种用于治疗肿瘤的基因工程细胞 |
| CA3107119A1 (en) * | 2018-07-31 | 2020-02-06 | Washington University | Use of interleukin-7 and chimeric antigen receptor (car)-bearing immune effector cells for treating tumor |
| EP3829605A4 (en) * | 2018-08-01 | 2022-10-19 | ImmunityBio, Inc. | QUADRICISTRONIC SYSTEM WITH A HOMING RECEPTOR OR A CYTOKINE AND A CHIMERIC ANTIGEN RECEPTOR FOR GENETIC MODIFICATION OF IMMUNOTHERAPIES |
| US20210308171A1 (en) * | 2018-08-07 | 2021-10-07 | The Broad Institute, Inc. | Methods for combinatorial screening and use of therapeutic targets thereof |
| GB201813178D0 (en) * | 2018-08-13 | 2018-09-26 | Autolus Ltd | Cell |
| US11142740B2 (en) | 2018-08-14 | 2021-10-12 | Inscripta, Inc. | Detection of nuclease edited sequences in automated modules and instruments |
| IL292273B2 (en) | 2018-08-14 | 2023-10-01 | Inscripta Inc | Devices, modules and methods for improved detection of edited sequences in living cells |
| CN109306016B (zh) * | 2018-08-15 | 2021-10-22 | 华东师范大学 | 共表达细胞因子il-7的nkg2d-car-t细胞及其用途 |
| US20220348682A1 (en) | 2018-08-30 | 2022-11-03 | Innovative Cellular Therapeutics Holdings, Ltd. | Chimeric antigen receptor cells for treating solid tumor |
| JP2021534783A (ja) | 2018-08-31 | 2021-12-16 | ノバルティス アーゲー | キメラ抗原受容体発現細胞を作製する方法 |
| US20210171909A1 (en) | 2018-08-31 | 2021-06-10 | Novartis Ag | Methods of making chimeric antigen receptor?expressing cells |
| AU2019344795A1 (en) * | 2018-09-17 | 2021-03-25 | The United States Of America, As Represented By The Secretary, Department Of Health And Human Services | Bicistronic chimeric antigen receptors targeting CD19 and CD20 and their uses |
| JP2022501067A (ja) * | 2018-09-26 | 2022-01-06 | 上海恒潤達生生物科技有限公司Hrain Biotechnology Co., Ltd. | Bcma及びcd19を標的とするキメラ抗原受容体、並びにその使用 |
| US20210338729A1 (en) * | 2018-10-12 | 2021-11-04 | Icell Gene Therapeutics Llc | CHIMERIC ANTIGEN RECEPTORS (CARs) COMPOSITIONS AND METHODS OF USE THEREOF |
| JP2022512766A (ja) * | 2018-10-17 | 2022-02-07 | センティ バイオサイエンシズ インコーポレイテッド | 組み合わせがん免疫療法 |
| US11214781B2 (en) | 2018-10-22 | 2022-01-04 | Inscripta, Inc. | Engineered enzyme |
| KR102533540B1 (ko) * | 2018-10-31 | 2023-05-17 | 난트퀘스트, 인크. | Pd-l1 키메라 항원 수용체-발현 nk 세포에 의한 pd-l1-양성 악성종양의 제거(elimination of pd-l1-positive malignancies by pd-l1 chimeric antigen receptor-expressing nk cells) |
| AU2019371340B2 (en) * | 2018-10-31 | 2022-03-03 | Nantkwest, Inc. | Elimination of CD19-positive lymphoid malignancies by CD19-CAR expressing NK cells |
| US10918667B2 (en) | 2018-11-20 | 2021-02-16 | Innovative Cellular Therapeutics CO., LTD. | Modified cell expressing therapeutic agent and uses thereof |
| JP2022513131A (ja) * | 2018-11-26 | 2022-02-07 | ンカルタ・インコーポレイテッド | 複数の免疫細胞タイプの同時増殖のための方法、関連する組成物および癌免疫療法におけるそれらの使用 |
| CA3177829A1 (en) | 2018-12-12 | 2020-06-18 | Kite Pharma, Inc. | Chimeric antigen and t cell receptors and methods of use |
| EP3908613A4 (en) * | 2019-01-07 | 2022-10-19 | Celluris Participações Ltda. | TANDEM BISPECIFIC CAR RECEPTOR AND METHOD FOR MODULATING A TUMOR MICROENVIRONMENT |
| WO2020172177A1 (en) * | 2019-02-18 | 2020-08-27 | Memorial Sloan-Kettering Cancer Center | Combinations of multiple chimeric antigen receptors for immunotherapy |
| SG11202107825WA (en) | 2019-02-25 | 2021-09-29 | Novartis Ag | Mesoporous silica particles compositions for viral delivery |
| CN113766956A (zh) | 2019-03-05 | 2021-12-07 | 恩卡尔塔公司 | Cd19定向性嵌合抗原受体及其在免疫疗法中的用途 |
| US20200283495A1 (en) * | 2019-03-08 | 2020-09-10 | ST Phi Therapeutics | Chimeric Endocytic Receptors and Method of Use Thereof |
| WO2020190902A1 (en) * | 2019-03-19 | 2020-09-24 | H. Lee Moffitt Cancer Center And Research Institute Inc. | Chimeric antigen receptors with enhanced tumor infiltration |
| US20230074800A1 (en) | 2019-03-21 | 2023-03-09 | Novartis Ag | Car-t cell therapies with enhanced efficacy |
| CN113631713A (zh) | 2019-03-25 | 2021-11-09 | 因思科瑞普特公司 | 酵母中的同时多重基因组编辑 |
| US11001831B2 (en) | 2019-03-25 | 2021-05-11 | Inscripta, Inc. | Simultaneous multiplex genome editing in yeast |
| EP3953455A1 (en) | 2019-04-12 | 2022-02-16 | Novartis AG | Methods of making chimeric antigen receptor-expressing cells |
| EP3959320A1 (en) | 2019-04-24 | 2022-03-02 | Novartis AG | Compositions and methods for selective protein degradation |
| WO2020219989A1 (en) * | 2019-04-26 | 2020-10-29 | Aleta Biotherapeutics Inc. | Compositions and methods for treatment of cancer |
| AU2020265679A1 (en) * | 2019-04-30 | 2021-12-23 | Senti Biosciences, Inc. | Chimeric receptors and methods of use thereof |
| US20200376032A1 (en) * | 2019-05-31 | 2020-12-03 | Case Western Reserve University | Targeting b cell activating factor receptor (baff-r) using ligand-based chimeric antigen receptor (car)-t cells |
| CA3139122C (en) | 2019-06-06 | 2023-04-25 | Inscripta, Inc. | Curing for recursive nucleic acid-guided cell editing |
| CN110452294B (zh) * | 2019-08-06 | 2020-08-07 | 复旦大学 | 五种铰链区及其嵌合抗原受体和免疫细胞 |
| US20210047423A1 (en) | 2019-08-16 | 2021-02-18 | Janssen Biotech, Inc. | Therapeutic immune cells with improved function and methods for making the same |
| CN112442509A (zh) * | 2019-08-29 | 2021-03-05 | 爱博赛特生物治疗公司 | Cd19-cd20双特异性和双通道car-t及其使用方法 |
| EP4028413A1 (en) | 2019-09-10 | 2022-07-20 | Obsidian Therapeutics, Inc. | Ca2-il15 fusion proteins for tunable regulation |
| WO2021102059A1 (en) | 2019-11-19 | 2021-05-27 | Inscripta, Inc. | Methods for increasing observed editing in bacteria |
| MX2022006391A (es) | 2019-11-26 | 2022-06-24 | Novartis Ag | Receptores de antigeno quimerico que se unen a bcma y cd19 y usos de los mismos. |
| EP4069748A4 (en) * | 2019-12-05 | 2023-11-15 | Vycellix, Inc. | MODULATORS OF THE IMMUNE ESCAPE MECHANISM FOR UNIVERSAL CELL THERAPY |
| JP2023507566A (ja) | 2019-12-18 | 2023-02-24 | インスクリプタ, インコーポレイテッド | 核酸誘導ヌクレアーゼ編集済み細胞のin vivo検出のためのカスケード/dCas3相補性アッセイ |
| CA3164666A1 (en) * | 2020-01-13 | 2021-07-22 | Kanya Lakshmi RAJANGAM | Bcma-directed cellular immunotherapy compositions and methods |
| AU2021213705A1 (en) | 2020-01-27 | 2022-06-16 | Inscripta, Inc. | Electroporation modules and instrumentation |
| US11230699B2 (en) | 2020-01-28 | 2022-01-25 | Immunitybio, Inc. | Chimeric antigen receptor-modified NK-92 cells targeting EGFR super-family receptors |
| US20210238535A1 (en) * | 2020-02-02 | 2021-08-05 | Inscripta, Inc. | Automated production of car-expressing cells |
| WO2021158783A1 (en) * | 2020-02-05 | 2021-08-12 | Washington University | Method of treating a solid tumor with a combination of an il-7 protein and car-bearing immune cells |
| CA3173737A1 (en) | 2020-02-27 | 2021-09-02 | Novartis Ag | Methods of making chimeric antigen receptor-expressing cells |
| IL295604A (en) | 2020-02-27 | 2022-10-01 | Novartis Ag | Methods for producing cells expressing a chimeric antigen receptor |
| WO2022074464A2 (en) | 2020-03-05 | 2022-04-14 | Neotx Therapeutics Ltd. | Methods and compositions for treating cancer with immune cells |
| US20210332388A1 (en) | 2020-04-24 | 2021-10-28 | Inscripta, Inc. | Compositions, methods, modules and instruments for automated nucleic acid-guided nuclease editing in mammalian cells |
| PE20230494A1 (es) | 2020-05-08 | 2023-03-23 | Alpine Immune Sciences Inc | Proteinas inmunomoduladoras inhibidoras de april y baff y metodos de uso de las mismas |
| US11787841B2 (en) | 2020-05-19 | 2023-10-17 | Inscripta, Inc. | Rationally-designed mutations to the thrA gene for enhanced lysine production in E. coli |
| CA3180750A1 (en) * | 2020-05-29 | 2021-12-02 | Yupo Ma | Engineered immune cells, compositions and methods thereof |
| AU2021288224A1 (en) | 2020-06-11 | 2023-01-05 | Novartis Ag | ZBTB32 inhibitors and uses thereof |
| EP4199960A2 (en) | 2020-08-21 | 2023-06-28 | Novartis AG | Compositions and methods for in vivo generation of car expressing cells |
| TW202216990A (zh) * | 2020-09-08 | 2022-05-01 | 中國大陸商亘喜生物科技(上海)有限公司 | 用於t細胞工程化的組合物和方法 |
| US11299731B1 (en) | 2020-09-15 | 2022-04-12 | Inscripta, Inc. | CRISPR editing to embed nucleic acid landing pads into genomes of live cells |
| WO2022076256A1 (en) * | 2020-10-05 | 2022-04-14 | Icell Gene Therapeutics Llc | Engineered immune cells for immunotherapy using endoplasmic retention techniques |
| US11512297B2 (en) | 2020-11-09 | 2022-11-29 | Inscripta, Inc. | Affinity tag for recombination protein recruitment |
| WO2022143928A1 (zh) * | 2020-12-31 | 2022-07-07 | 亘喜生物科技(上海)有限公司 | 膜融合蛋白及其在免疫细胞中的应用 |
| CA3204158A1 (en) | 2021-01-04 | 2022-07-07 | Juhan Kim | Mad nucleases |
| WO2022150269A1 (en) | 2021-01-07 | 2022-07-14 | Inscripta, Inc. | Mad nucleases |
| CN112778427B (zh) * | 2021-01-29 | 2022-03-15 | 武汉思安医疗技术有限公司 | 双特异性cs1-bcma car-t细胞及其应用 |
| US11884924B2 (en) | 2021-02-16 | 2024-01-30 | Inscripta, Inc. | Dual strand nucleic acid-guided nickase editing |
| CN112698037B (zh) * | 2021-03-25 | 2021-06-25 | 北京积水潭医院 | 一种检测多发性骨髓瘤治疗效果的抗体组合物及其试剂盒和应用 |
| EP4330381A1 (en) | 2021-04-27 | 2024-03-06 | Novartis AG | Viral vector production system |
| AU2022330406A1 (en) | 2021-08-20 | 2024-03-07 | Novartis Ag | Methods of making chimeric antigen receptor–expressing cells |
| CN114835820B (zh) * | 2022-04-14 | 2023-01-06 | 呈诺再生医学科技(珠海横琴新区)有限公司 | 用于基因改造的多能干细胞及、自然杀伤细胞的嵌合型Fc受体 |
| CN115232217B (zh) * | 2022-07-04 | 2023-06-06 | 深圳市先康达生命科学有限公司 | 一种SynNotch结构及其应用 |
| GB202209920D0 (en) * | 2022-07-06 | 2022-08-17 | Autolus Ltd | Cell |
Family Cites Families (64)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US6764681B2 (en) | 1991-10-07 | 2004-07-20 | Biogen, Inc. | Method of prophylaxis or treatment of antigen presenting cell driven skin conditions using inhibitors of the CD2/LFA-3 interaction |
| WO2001029058A1 (en) | 1999-10-15 | 2001-04-26 | University Of Massachusetts | Rna interference pathway genes as tools for targeted genetic interference |
| US6326193B1 (en) | 1999-11-05 | 2001-12-04 | Cambria Biosciences, Llc | Insect control agent |
| WO2001096584A2 (en) | 2000-06-12 | 2001-12-20 | Akkadix Corporation | Materials and methods for the control of nematodes |
| US20030147865A1 (en) | 2002-02-07 | 2003-08-07 | Benoit Salomon | Cell therapy using immunoregulatory T-cells |
| US20080254027A1 (en) | 2002-03-01 | 2008-10-16 | Bernett Matthew J | Optimized CD5 antibodies and methods of using the same |
| WO2004022097A1 (en) | 2002-09-05 | 2004-03-18 | Medimmune, Inc. | Methods of preventing or treating cell malignancies by administering cd2 antagonists |
| US7435596B2 (en) | 2004-11-04 | 2008-10-14 | St. Jude Children's Research Hospital, Inc. | Modified cell line and method for expansion of NK cell |
| WO2005108415A2 (en) | 2004-04-30 | 2005-11-17 | Biogen Idec Ma Inc. | Membrane associated molecules |
| US20050277587A1 (en) | 2004-06-10 | 2005-12-15 | Academia Sinica | CD7 as biomarker and therapeutic target for psoriasis |
| EP1777294A1 (en) * | 2005-10-20 | 2007-04-25 | Institut National De La Sante Et De La Recherche Medicale (Inserm) | IL-15Ralpha sushi domain as a selective and potent enhancer of IL-15 action through IL-15Rbeta/gamma, and hyperagonist (IL15Ralpha sushi -IL15) fusion proteins |
| EP2441846A3 (en) | 2006-01-09 | 2012-07-25 | The Regents Of the University of California | Immunostimulatory combinations of TNFRSF, TLR, NLR, RHR, purinergic receptor, and cytokine receptor agoinsts for vaccines and tumor immunotherapy |
| CA2655471C (en) | 2006-06-12 | 2017-02-14 | Centocor, Inc. | Method for detecting il-16 activity and modulation of il-16 activity based on rantes proxy levels |
| EP3284825B1 (en) | 2006-11-02 | 2021-04-07 | Biomolecular Holdings LLC | Methods of producing hybrid polypeptides with moving parts |
| WO2009091826A2 (en) | 2008-01-14 | 2009-07-23 | The Board Of Regents Of The University Of Texas System | Compositions and methods related to a human cd19-specific chimeric antigen receptor (h-car) |
| EP2389443B1 (en) | 2009-01-23 | 2018-11-14 | Roger Williams Hospital | Retroviral vectors encoding multiple highly homologous non-viral polypeptides and the use of same |
| US20120058082A1 (en) | 2009-05-13 | 2012-03-08 | Genzyme Corporation | Methods and compositions for treatment |
| EP3345620A1 (en) | 2009-05-13 | 2018-07-11 | Genzyme Corporation | Methods and compositions for treating lupus |
| EP2325322A1 (en) | 2009-11-23 | 2011-05-25 | 4-Antibody AG | Retroviral vector particles and methods for their generation and use |
| SG10201510092QA (en) | 2010-12-09 | 2016-01-28 | Univ Pennsylvania | Use of chimeric antigen receptor-modified t cells to treat cancer |
| US9233125B2 (en) | 2010-12-14 | 2016-01-12 | University Of Maryland, Baltimore | Universal anti-tag chimeric antigen receptor-expressing T cells and methods of treating cancer |
| CA2832356C (en) | 2011-04-06 | 2017-12-19 | Sanbio, Inc. | Methods and compositions for modulating peripheral immune function |
| JP6159724B2 (ja) | 2011-08-23 | 2017-07-05 | ロシュ グリクアート アーゲー | T細胞活性化抗原に対して特異的な二重特異性抗体及び腫瘍抗原および使用方法 |
| EP3594245A1 (en) * | 2012-02-13 | 2020-01-15 | Seattle Children's Hospital d/b/a Seattle Children's Research Institute | Bispecific chimeric antigen receptors and therapeutic uses thereof |
| MX2014010183A (es) | 2012-02-22 | 2015-03-20 | Univ Pennsylvania | Composiciones y metodos para generar una poblacion persistente de celulas t utiles para el tratamiento de cancer. |
| TWI619729B (zh) | 2012-04-02 | 2018-04-01 | 再生元醫藥公司 | 抗-hla-b*27抗體及其用途 |
| MX2015000428A (es) * | 2012-07-13 | 2015-03-12 | Univ Pennsylvania | Composiciones y metodos para regular celulas t car. |
| SG11201502598SA (en) | 2012-10-02 | 2015-05-28 | Sloan Kettering Inst Cancer | Compositions and methods for immunotherapy |
| WO2014055657A1 (en) * | 2012-10-05 | 2014-04-10 | The Trustees Of The University Of Pennsylvania | Use of a trans-signaling approach in chimeric antigen receptors |
| AU2013204922B2 (en) | 2012-12-20 | 2015-05-14 | Celgene Corporation | Chimeric antigen receptors |
| KR20220100093A (ko) * | 2013-02-06 | 2022-07-14 | 안트로제네시스 코포레이션 | 개선된 특이성을 갖는 변경된 t 림프구 |
| CN105142677B (zh) | 2013-02-15 | 2019-08-30 | 加利福尼亚大学董事会 | 嵌合抗原受体及其使用方法 |
| US9745368B2 (en) * | 2013-03-15 | 2017-08-29 | The Trustees Of The University Of Pennsylvania | Targeting cytotoxic cells with chimeric receptors for adoptive immunotherapy |
| PT2997141T (pt) | 2013-05-13 | 2022-11-23 | Cellectis | Recetor de antigénio quimérico específico para cd19 e utilizações do mesmo |
| WO2014186469A2 (en) * | 2013-05-14 | 2014-11-20 | Board Of Regents, The University Of Texas System | Human application of engineered chimeric antigen receptor (car) t-cells |
| CA2920539C (en) | 2013-08-08 | 2024-01-02 | Cytune Pharma | Combined pharmaceutical composition |
| SG11201603228TA (en) | 2013-10-31 | 2016-05-30 | Hutchinson Fred Cancer Res | Modified hematopoietic stem/progenitor and non-t effector cells, and uses thereof |
| US9493563B2 (en) | 2013-11-04 | 2016-11-15 | Glenmark Pharmaceuticals S.A. | Production of T cell retargeting hetero-dimeric immunoglobulins |
| PT3071222T (pt) | 2013-11-21 | 2020-11-20 | Ucl Business Plc | Célula |
| ES2963718T3 (es) | 2014-01-21 | 2024-04-01 | Novartis Ag | Capacidad presentadora de antígenos de células CAR-T potenciada mediante introducción conjunta de moléculas co-estimuladoras |
| WO2015120180A1 (en) | 2014-02-05 | 2015-08-13 | The University Of Chicago | Chimeric antigen receptors recognizing cancer-specific tn glycopeptide variants |
| MX2016010345A (es) | 2014-02-14 | 2017-01-23 | Cellectis | Celulas para inmunoterapia diseñadas para dirigir antigeno presente tanto en celulas inmunes y celulas patologicas. |
| US20170029774A1 (en) | 2014-04-10 | 2017-02-02 | Seattle Children's Hospital (dba Seattle Children' Research Institute) | Production of engineered t-cells by sleeping beauty transposon coupled with methotrexate selection |
| CN113604491A (zh) | 2014-05-02 | 2021-11-05 | 宾夕法尼亚大学董事会 | 嵌合自身抗体受体t细胞的组合物和方法 |
| GB2541599B (en) | 2014-05-14 | 2020-05-20 | Carsgen Therapeutics Ltd | Nucleic acid for coding chimeric antigen receptor protein and T lymphocyte for expression of chimeric antigen receptor protein |
| US11542488B2 (en) | 2014-07-21 | 2023-01-03 | Novartis Ag | Sortase synthesized chimeric antigen receptors |
| TW202140557A (zh) * | 2014-08-19 | 2021-11-01 | 瑞士商諾華公司 | 使用cd123嵌合抗原受體治療癌症 |
| US20170260261A1 (en) | 2014-08-28 | 2017-09-14 | Bioatla, Llc | Conditionally Active Chimeric Antigen Receptors for Modified T-Cells |
| CA2963327A1 (en) * | 2014-10-07 | 2016-04-14 | Cellectis | Method for modulating car-induced immune cells activity |
| CN107002045A (zh) | 2014-12-24 | 2017-08-01 | Ucl商务股份有限公司 | 细胞 |
| CN107249602B (zh) * | 2015-02-27 | 2021-09-28 | 美商生物细胞基因治疗有限公司 | 靶向血液恶性肿瘤之嵌合抗原受体(car)、其组合物及使用方法 |
| LT3280729T (lt) * | 2015-04-08 | 2022-08-10 | Novartis Ag | Terapijos cd20, terapijos cd22 ir kombinuotos terapijos su cd19 chimerinį antigeno receptorių (car) ekspresuojančia ląstele |
| US20190135894A1 (en) * | 2015-06-25 | 2019-05-09 | iCell Gene Therapeuticics LLC | COMPOUND CHIMERIC ANTIGEN RECEPTOR (cCAR) TARGETING MULTIPLE ANTIGENS, COMPOSITIONS AND METHODS OF USE THEREOF |
| JP6961497B2 (ja) | 2015-06-25 | 2021-11-05 | アイセル・ジーン・セラピューティクス・エルエルシー | キメラ抗体受容体(CARs)の構成およびその使用方法 |
| US11173179B2 (en) * | 2015-06-25 | 2021-11-16 | Icell Gene Therapeutics Llc | Chimeric antigen receptor (CAR) targeting multiple antigens, compositions and methods of use thereof |
| US20220241327A1 (en) * | 2015-06-25 | 2022-08-04 | Icell Gene Therapeutics Llc | Chimeric Antigen Receptor (CAR) Targeting Multiple Antigens, Compositions and Methods of Use Thereof |
| CN105384825B (zh) * | 2015-08-11 | 2018-06-01 | 南京传奇生物科技有限公司 | 一种基于单域抗体的双特异性嵌合抗原受体及其应用 |
| CN105087495B (zh) * | 2015-08-21 | 2016-04-27 | 深圳市茵冠生物科技有限公司 | 双嵌合抗原受体修饰的t淋巴细胞及其制备方法 |
| GB201518817D0 (en) | 2015-10-23 | 2015-12-09 | Autolus Ltd | Cell |
| US20180371052A1 (en) * | 2015-12-22 | 2018-12-27 | Icell Gene Therapeutics Llc | Chimeric antigen receptors and enhancement of anti-tumor activity |
| EP3474867A4 (en) * | 2016-06-24 | 2020-05-20 | iCell Gene Therapeutics LLC | CHIMERIC ANTIGEN (CAR) RECEPTORS, COMPOSITIONS AND RELATED METHODS |
| US20200223918A1 (en) * | 2017-06-21 | 2020-07-16 | Icell Gene Therapeutics Llc | CHIMERIC ANTIGEN RECEPTORS (CARs), COMPOSITIONS AND METHODS THEREOF |
| EP3694886A4 (en) * | 2017-10-12 | 2021-09-22 | iCell Gene Therapeutics, LLC | COMPOSITE CHIMERIC ANTIGEN RECEPTOR (CCAR) TARGETING MULTIPLE ANTIGENS, COMPOSITIONS AND METHOD OF USING THEREOF |
| MX2020005651A (es) * | 2017-11-30 | 2020-10-28 | Novartis Ag | Receptor de antigeno quimerico dirigido a bcma y usos del mismo. |
-
2016
- 2016-06-24 JP JP2017567178A patent/JP6961497B2/ja active Active
- 2016-06-24 SG SG10201913682QA patent/SG10201913682QA/en unknown
- 2016-06-24 CN CN201680036459.4A patent/CN107849112B/zh active Active
- 2016-06-24 EP EP16815399.7A patent/EP3313872A4/en active Pending
- 2016-06-24 CA CA2990177A patent/CA2990177A1/en active Pending
- 2016-06-24 WO PCT/US2016/039306 patent/WO2016210293A1/en active Application Filing
- 2016-06-24 AU AU2016283102A patent/AU2016283102B2/en active Active
- 2016-06-24 KR KR1020187002389A patent/KR20180021137A/ko not_active Application Discontinuation
- 2016-06-24 CN CN202210523997.3A patent/CN115058395A/zh active Pending
- 2016-06-24 US US15/739,596 patent/US11655452B2/en active Active
-
2017
- 2017-12-19 IL IL256410A patent/IL256410B/en unknown
-
2021
- 2021-05-28 AU AU2021203481A patent/AU2021203481B2/en active Active
- 2021-10-12 JP JP2021167360A patent/JP7254870B2/ja active Active
- 2021-10-31 IL IL287718A patent/IL287718A/en unknown
-
2023
- 2023-03-29 JP JP2023053851A patent/JP2023076570A/ja active Pending
- 2023-09-22 AU AU2023233207A patent/AU2023233207A1/en active Pending
Cited By (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2021225399A1 (ko) * | 2020-05-08 | 2021-11-11 | (주)에스엠티바이오 | 암 치료를 위한 키메라 항원 수용체 |
Also Published As
| Publication number | Publication date |
|---|---|
| JP2022001067A (ja) | 2022-01-06 |
| CN107849112B (zh) | 2022-04-01 |
| JP2018518974A (ja) | 2018-07-19 |
| CN115058395A (zh) | 2022-09-16 |
| US11655452B2 (en) | 2023-05-23 |
| AU2023233207A1 (en) | 2023-10-12 |
| JP2023076570A (ja) | 2023-06-01 |
| IL287718A (en) | 2021-12-01 |
| JP6961497B2 (ja) | 2021-11-05 |
| EP3313872A1 (en) | 2018-05-02 |
| SG10201913682QA (en) | 2020-03-30 |
| WO2016210293A1 (en) | 2016-12-29 |
| CN107849112A (zh) | 2018-03-27 |
| IL256410B (en) | 2022-01-01 |
| US20180187149A1 (en) | 2018-07-05 |
| AU2016283102A1 (en) | 2018-01-18 |
| IL256410A (en) | 2018-02-28 |
| TW201713768A (zh) | 2017-04-16 |
| JP7254870B2 (ja) | 2023-04-10 |
| AU2016283102B2 (en) | 2021-03-11 |
| EP3313872A4 (en) | 2019-03-20 |
| AU2021203481A1 (en) | 2021-07-01 |
| AU2021203481B2 (en) | 2023-08-03 |
| CA2990177A1 (en) | 2016-12-29 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP7254870B2 (ja) | キメラ抗体受容体(CARs)の構成およびその使用方法 | |
| US20230340113A1 (en) | CHIMERIC ANTIGEN RECEPTORS (CARs), COMPOSITIONS AND METHODS THEREOF | |
| JP7379399B2 (ja) | 血液系腫瘍を標的としたキメラ抗体受容体(CARs)の構成およびその使用方法 | |
| US20200223918A1 (en) | CHIMERIC ANTIGEN RECEPTORS (CARs), COMPOSITIONS AND METHODS THEREOF | |
| CN117721084A (zh) | 靶向多种抗原的复合嵌合抗原受体(cCAR)及其组成和使用方法 | |
| CN113286874A (zh) | 嵌合抗原受体(CARs)组合物及使用方法 | |
| TWI833684B (zh) | 嵌合抗原受體(car)、組合物及其使用方法 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A201 | Request for examination | ||
| E902 | Notification of reason for refusal |