KR20130066632A - 치료적 저밀도 지단백질-관련 단백질 6 (lrp6) 항체에 대한 조성물 및 사용 방법 - Google Patents
치료적 저밀도 지단백질-관련 단백질 6 (lrp6) 항체에 대한 조성물 및 사용 방법 Download PDFInfo
- Publication number
- KR20130066632A KR20130066632A KR1020127031870A KR20127031870A KR20130066632A KR 20130066632 A KR20130066632 A KR 20130066632A KR 1020127031870 A KR1020127031870 A KR 1020127031870A KR 20127031870 A KR20127031870 A KR 20127031870A KR 20130066632 A KR20130066632 A KR 20130066632A
- Authority
- KR
- South Korea
- Prior art keywords
- seq
- antibody
- lrp6
- fragment
- monovalent
- Prior art date
Links
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/2863—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against receptors for growth factors, growth regulators
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/395—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/46—Hybrid immunoglobulins
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/505—Medicinal preparations containing antigens or antibodies comprising antibodies
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/20—Immunoglobulins specific features characterized by taxonomic origin
- C07K2317/21—Immunoglobulins specific features characterized by taxonomic origin from primates, e.g. man
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/30—Immunoglobulins specific features characterized by aspects of specificity or valency
- C07K2317/31—Immunoglobulins specific features characterized by aspects of specificity or valency multispecific
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/30—Immunoglobulins specific features characterized by aspects of specificity or valency
- C07K2317/33—Crossreactivity, e.g. for species or epitope, or lack of said crossreactivity
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/30—Immunoglobulins specific features characterized by aspects of specificity or valency
- C07K2317/34—Identification of a linear epitope shorter than 20 amino acid residues or of a conformational epitope defined by amino acid residues
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/30—Immunoglobulins specific features characterized by aspects of specificity or valency
- C07K2317/35—Valency
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/55—Fab or Fab'
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/60—Immunoglobulins specific features characterized by non-natural combinations of immunoglobulin fragments
- C07K2317/62—Immunoglobulins specific features characterized by non-natural combinations of immunoglobulin fragments comprising only variable region components
- C07K2317/622—Single chain antibody (scFv)
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/60—Immunoglobulins specific features characterized by non-natural combinations of immunoglobulin fragments
- C07K2317/64—Immunoglobulins specific features characterized by non-natural combinations of immunoglobulin fragments comprising a combination of variable region and constant region components
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/76—Antagonist effect on antigen, e.g. neutralization or inhibition of binding
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/90—Immunoglobulins specific features characterized by (pharmaco)kinetic aspects or by stability of the immunoglobulin
- C07K2317/92—Affinity (KD), association rate (Ka), dissociation rate (Kd) or EC50 value
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/90—Immunoglobulins specific features characterized by (pharmaco)kinetic aspects or by stability of the immunoglobulin
- C07K2317/94—Stability, e.g. half-life, pH, temperature or enzyme-resistance
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
Abstract
본 발명은 LRP6을 표적으로 하는 항체, 및 그의 조성물 및 사용 방법에 관한 것이다.
Description
관련 출원
본원은 그 내용 전체가 본원에 참고로 포함되는, 2010년 5월 6일 출원된 미국 특허 가출원 일련 번호 61/331,985를 기초로 한 우선권을 주장한다.
발명의 분야
본 발명은 LRP6에 특이적으로 결합하는 항체에 관한 것이다. 본 발명은 보다 구체적으로 LRP6 길항제인 특이적 항체에 관한 것이다.
Wnt/β-카테닌 경로는 β-카테닌의 단백질 안정성의 조정을 통해 발생 및 조직 항상성 동안 다양한 생물학적 과정을 조절한다 ([Clevers et al., (2006) Cell 127:469-480]; 및 [Logan et al., (2004) Annu. Rev Cell Dev. Biol 20:781-810]). Wnt 신호 전달의 부재 하에, 세포질 β-카테닌은 선종성 결장 폴립증 (APC), 액신 (Axin), 및 글리코겐 합성효소 키나제 3 (GSK3)을 비롯한 다수의 단백질을 함유하는 β-카테닌 파괴 복합체와 연합된다. 상기 복합체에서, β-카테닌은 GSK3에 의해 구성적으로 인산화되고, 프로테아솜 경로에 의해 파괴된다. Wnt 신호는 2개의 구분되는 수용체 (뱀모양 (serpentine) 수용체 프리즐드 (Frizzled), 및 단일-막횡단 단백질 LRP5 또는 LRP6)를 통해 혈장 막을 가로질러 도입된다. Wnt 단백질은 프리즐드-LRP5/6 신호 전달 복합체의 조립을 촉진하고, GSK3 및 카제인 키나제 I에 의한 LRP5/6의 세포질 PPPSPxS 모티프의 인산화를 유도한다. 인산화된 LRP5/6은 액신에 결합하고, β-카테닌 분해 복합체를 불활성화한다. 안정화된 β-카테닌은 핵으로 들어와서, TCF 패밀리 전사 인자에 결합하고, 전사를 일으킨다.
LRP5/6의 큰 세포외 도메인은 4개의 YWTD-형 β-프로펠러 (propeller) 영역을 함유하고, 그 각각은 EGF-유사 도메인 및 LDLR 도메인으로 이어진다. 각각의 프로펠러 영역은 6-날개깃의 β-프로펠러 구조를 형성하는 6개의 YWTD 모티프를 함유한다. 생화학적 연구에서는 Wnt 단백질이 프리즐드 및 LRP6 모두와 물리적으로 상호작용하여, 프리즐드-LRP6 신호 전달 복합체의 형성을 유도함을 제안하였다 ([Semenov et al., (2001) Curr. Biol 11, 951-961]; 및 [Tamai et al., (2000) Nature 407, 530-535]). Wnt 단백질 외에, LRP5/6의 큰 세포외 도메인은 Wnt 길항제, DKK1 및 스클레로스틴 (Sclerostin) (SOST), 및 Wnt 효능제 R-스폰딘 (Spondin)을 비롯한 다수의 분비형 Wnt 조절물질 (modulator)에 결합한다.
Wnt 신호 전달 경로의 탈조절은 많은 인간 질환과 연관되었다. Wnt/LRP5/6 신호 전달 경로는 조직 항상성 및 재생에서 중요한 역할을 수행한다. Wnt 신호 전달은 골모세포의 성장 및 분화를 증가시킴으로써 뼈 형성을 촉진한다 (Baron et al., (2006) Curr. Top. Dev. Biol 76:103-127). LRP5의 기능 획득 (Gain-of-function) 돌연변이 ([Boyden et al., (2002) N. Engl. J Med 346:1513-1521]; [Little et al., (2002) Am. J Hum. Genet. 70:11-19]; [Van Wesenbeeck et al., (2003) Am. J Hum. Genet. 72:763-71]) 및 Wnt 길항제 SOST의 기능 상실 (loss-of-function) 돌연변이 ([Balemans et al., (2001) Hum. Mol Genet. 10:537-543]; [Brunkow et al., (2001) Am. J Hum. Genet. 68:577-589]) 둘 모두는 높은 골 질량 질환을 유발한다. 또한, Wnt 신호 전달은 창자샘 (intestinal crypt)에서 줄기 세포의 증식 상태를 유지함으로써 장 상피의 항상성을 위해 매우 중요하다 (Pinto et al., (2005) Biol Cell 97:185-196). 또한, Wnt 신호 전달은 신장 복구 및 재생에서 중요하다 (Lin SL PNAS 107:4194, 2010). 또한, APC 및 β-카테닌과 같은 경로 성분에서의 돌연변이는 인간 암과 연관되었다. 최근의 연구에서는 Wnt 단백질의 과발현 및/또는 Wnt 길항제, 예컨대 DKK1, WISP 및 sFRP의 침묵이 암 발생 및 진행을 촉진함을 제안한다 ([Akiri et al., (2009) Oncogene 28:2163-2172]; [Bafico et al., (2004) Cancer Cell 6:497-506]; [Suzuki et al., (2004) Nat Genet. 36:417-422]; [Taniguchi et al., (2005) Oncogene. 24:7946-7952]; [Veeck et al., (2006) Oncogene. 25:3479-3488]; [Zeng et al., (2007) Hum. Pathol. 38:120-133]). 또한, Wnt 신호 전달은 암 줄기 세포의 유지에 연루되었다 ([Jamieson et al., (2004) Cancer Cell 6:531-533] 및 [Zhao et al., (2007) Cancer Cell 12:528-541]).
따라서, 이상 정형적 (canonical) Wnt 신호 전달과 연관된 질환에 대한 요법으로서 세포외 수준에서 Wnt 신호 전달을 길항하는 작용제에 대한 필요성이 존재한다.
발명의 개요
본 발명은 정형적 Wnt 신호 전달 경로를 억제하거나 향상시키는 LRP6 항체 (예를 들어, 일가, 이가) 및 LRP6 항체의 제조 방법을 제공한다. 본 발명의 LRP6 항체는 구분되는 LRP6 β-프로펠러 영역에 결합한다. 프로펠러 1 항체는 β-프로펠러 1 도메인에 결합하고 프로펠러 1-의존성 Wnt, 예컨대 Wnt1, Wnt2, Wnt6, Wnt7A, Wnt7B, Wnt9, Wnt10A, Wnt10B를 차단한다. 프로펠러 3 항체는 β-프로펠러 3 도메인에 결합하고, 프로펠러 3-의존성 Wnt, 예컨대 Wnt3a 및 Wnt3을 차단한다. 본 발명은 LRP6 항체가 프로펠러 1 및 프로펠러 3 리간드를 2개의 별개의 클래스로 분화시키고, LRP6 표적의 구분되는 에피토프에 결합한다는 놀라운 발견을 기초로 한다. 또 다른 놀라운 발견은 LRP6 항체 단편 (예를 들어, Fab)의 전장 IgG 항체로의 전환이 또 다른 단백질, 예컨대 Wnt1 또는 Wnt3 리간드의 존재 하에 Wnt 신호를 강화 (향상)하는 항체를 생성한다는 것이다. Wnt 리간드 이외에, LRP6 프로펠러 1 항체는 다른 프로펠러 1 결합 리간드 (예를 들어 스클레로스틴, Dkk1)와의 상호작용을 억제할 것으로 예상된다. 이와 유사하게, 프로펠러 3 항체는 다른 프로펠러 3 결합 리간드 (예를 들어, Dkk1)와의 상호작용을 억제할 것으로 예상된다. 또한, 프로펠러 1 및 3 결합 항체는 다른 Wnt 신호 전달 조절물질, 예를 들어 R-스폰딘의 활성에 영향을 줄 것으로 예상할 수 있다.
따라서, 한 측면에서, 본 발명은 차단되지 않은 저밀도 지단백질-관련 단백질 6 (LRP6) 결합 단백질의 존재 하에 하나 이상의 LRP6 수용체를 한데 모음으로써 Wnt 신호를 강화하는 LRP6에 대한 단리된 이가 항체 또는 그의 이가 단편에 관한 것이다. 한 실시양태에서, 항체는 프로펠러 1 및 프로펠러 3으로 이루어진 군 중에서 선택된 프로펠러 영역에 결합함으로써 하나 이상의 LRP6 수용체를 한데 모은다. 한 실시양태에서, LRP6 결합 단백질은 Wnt 1, Wnt 3 및 Wnt 3a로 이루어진 군 중에서 선택된 Wnt 결합 단백질이다.
또 다른 측면에서, 본 발명은 차단되지 않은 저밀도 지단백질-관련 단백질 6 (LRP6) 결합 단백질의 존재 하에 하나 이상의 LRP6 수용체를 한데 모음으로써 Wnt 신호의 강화를 방지하는 LRP6에 대한 단리된 이가 항체 또는 그의 이가 단편에 관한 것이다.
또 다른 측면에서, 본 발명은 차단되지 않은 저밀도 지단백질-관련 단백질 6 (LRP6) 결합 단백질의 존재 하에 하나 이상의 LRP6 수용체를 한데 모음으로써 Wnt 신호의 강화를 방지하는 LRP6에 대한 단리된 일가 항체 또는 그의 일가 단편에 관한 것이다.
한 측면에서, 본 발명은 해리 속도 상수 (KD)가 적어도 1 x 107 M-1, 108 M-1, 109 M-1, 1010 M-1, 또는 1011 M-1, 1012 M-1, 1013 M-1인, 저밀도 지단백질-관련 단백질 6 (LRP6) 단백질에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다. 한 실시양태에서, 항체 또는 그의 단편은 용액 평형 적정 검정에서 인간 LRP6에 대한 시험관내 결합에 의해 측정시에 정형적 Wnt 경로를 0.001 nM-1 μM의 KD로 억제하고, 여기서 항체 또는 그의 단편이 일가 또는 이가이다.
또 다른 측면에서, 본 발명은 용액 평형 적정 (SET) 검정에서 인간 저밀도 지단백질-관련 단백질 6 (LRP6)에 대한 시험관내 결합에 의해 측정시에 300 μM (0.3 pM) 이하의 EC50으로 정형적 Wnt 경로를 억제하는, LRP6 단백질에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편이 일가 또는 이가이다.
또 다른 측면에서, 본 발명은 차단되지 않은 저밀도 지단백질-관련 단백질 6 (LRP6) 결합 단백질의 존재 하에 하나 이상의 LRP6 수용체를 한데 모음으로써 Wnt 신호를 강화하는 LRP6에 대한 단리된 이가 항체 또는 그의 이가 단편에 관한 것이다. 항체는 프로펠러 1 및 프로펠러 3으로 이루어진 군 중에서 선택된 LRP6 분자의 프로펠러 영역에 결합함으로써 하나 이상의 LRP6 수용체를 한데 모은다. 한 실시양태에서, LRP6 결합 단백질은 Wnt1, Wnt3, 및 Wnt3a로 이루어진 군 중에서 선택된 Wnt 결합 단백질이다.
또 다른 측면에서, 본 발명은 표 1에 기재된 항체와 교차경쟁하는, LRP6 단백질에 대한 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편는 일가 또는 이가이고; 항체 또는 그의 단편은 표 1에 기재된 항체와 동일한 에피토프와 상호작용한다 (예를 들어, 결합, 입체 장애, 안정화/탈안정화, 공간 분포에 의해). 한 실시양태에서, 항체 또는 그의 단편은 모노클로날 항체이다. 또 다른 실시양태에서, 항체 또는 그의 단편은 인간 또는 인간화 항체이다. 또 다른 실시양태에서, 항체 또는 그의 단편은 키메라 항체이다. 한 실시양태에서, 항체 또는 그의 단편은 인간 중쇄 불변 영역 및 인간 경쇄 불변 영역을 포함한다. 한 실시양태에서, 항체 또는 그의 단편은 단일 쇄 항체이다. 또 다른 실시양태에서, 항체 또는 그의 단편은 Fab 단편이다. 또 다른 실시양태에서, 항체 또는 그의 단편은 scFv이다. 한 실시양태에서, 항체 또는 그의 단편은 인간 LRP6 및 시노몰구스 LRP6 둘 모두에 결합한다. 한 실시양태에서, 항체 또는 그의 단편은 IgG 이소형이다. 또 다른 실시양태에서, 항체 또는 그의 단편은, 아미노산이 각각의 인간 VH 또는 VL 배선 서열로부터의 항체 프레임워크로 치환된 프레임워크를 포함한다.
한 측면에서, 본 발명은 표 1 내의 임의의 항체의 1, 2, 3, 4, 5, 또는 6개의 CDR을 포함하는, LRP6에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본원에 개시된 LRP6 항체는 LRP6의 프로펠러 1 영역에 결합한다. 따라서, 본 발명은 서열 3, 서열 25, 서열 및 서열 43으로 이루어진 군 중에서 선택된 중쇄 CDR3을 포함하는, LRP6에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 14, 서열 34, 서열 36, 서열 44, 서열 60, 및 서열 62로 이루어진 군 중에서 선택된 중쇄 CDR1 및 서열 13, 서열 33, 서열 35, 서열 43, 서열 59, 및 서열 61로 이루어진 군 중에서 선택된 VL을 포함하는, LRP6에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 14를 포함하는 VH 및 서열 13을 포함하는 VL을 포함하는, LRP6에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 34를 포함하는 VH 및 서열 33을 포함하는 VL을 포함하는, LRP6에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 36을 포함하는 VH 및 서열 35를 포함하는 VL을 포함하는, LRP6에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 44를 포함하는 VH 및 서열 43을 포함하는 VL을 포함하는, LRP6에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 36을 포함하는 VH 및 서열 35를 포함하는 VL을 포함하는, LRP6에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 60을 포함하는 VH 및 서열 59를 포함하는 VL을 포함하는, LRP6에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 61을 포함하는 VH 및 서열 62를 포함하는 VL을 포함하는, LRP6에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 1, 2, 3, 21, 22, 23, 47, 48, 및 49에 동일한 적어도 하나의 중쇄 CDR 서열을 포함하는, 인간 LRP6 단백질에 결합하는 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 4, 5, 6, 24, 25, 26, 50, 51 및 52에 동일한 적어도 하나의 경쇄 CDR 서열을 포함하는, 인간 LRP6 단백질에 결합하는 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 1, 2, 3, 21, 22, 23, 47, 48, 및 49에 대해 적어도 95%의 서열 동일성을 갖는 적어도 하나의 중쇄 CDR 서열을 포함하는, 인간 LRP6 단백질에 결합하는 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 4, 5, 6, 24, 25, 26, 50, 51 및 52에 대해 적어도 95%의 서열 동일성을 갖는 적어도 하나의 경쇄 CDR 서열을 포함하는, 인간 LRP6 단백질에 결합하는 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 1의 중쇄 가변 영역 CDR1; 서열 2의 중쇄 가변 영역 CDR2; 서열 3의 중쇄 가변 영역 CDR3; 서열 4의 경쇄 가변 영역 CDR1; 서열 5의 경쇄 가변 영역 CDR2; 및 서열 6의 경쇄 가변 영역 CDR3을 포함하는 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 21의 중쇄 가변 영역 CDR1; 서열 22의 중쇄 가변 영역 CDR2; 서열 23의 중쇄 가변 영역 CDR3; 서열 24의 경쇄 가변 영역 CDR1; 서열 25의 경쇄 가변 영역 CDR2; 및 서열 26의 경쇄 가변 영역 CDR3을 포함하는 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 47의 중쇄 가변 영역 CDR1; 서열 48의 중쇄 가변 영역 CDR2; 서열 49의 중쇄 가변 영역 CDR3; 서열 50의 경쇄 가변 영역 CDR1; 서열 51의 경쇄 가변 영역 CDR2; 및 서열 52의 경쇄 가변 영역 CDR3을 포함하는 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 실시양태에서, LRP6에 결합하는 항체의 단편은 Fab, F(ab2)', F(ab)2', scFv, VHH, VH, VL, dAb로 이루어지는 군 중에서 선택되고, 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 항체 또는 그의 단편은 일가 또는 이가인 항체 또는 그의 단편 및 제약상 허용되는 담체를 포함하는 제약 조성물에 관한 것이다. 한 측면에서, 본 발명은 서열 14, 34, 36, 44, 60, 및 62에 대한 중쇄 가변 영역 동일성을 포함하는 폴리펩티드를 코딩하는 뉴클레오티드 서열을 포함하는 핵산; 서열 13, 33, 35, 43, 59 및 61에 대한 경쇄 가변 영역 동일성을 포함하는 폴리펩티드를 코딩하는 뉴클레오티드 서열을 포함하는 핵산; 서열 14, 34, 36, 44, 60, 및 62에 대해 적어도 98%의 서열 동일성을 갖는 중쇄 가변 영역을 포함하는 폴리펩티드를 코딩하는 뉴클레오티드 서열을 포함하는 핵산; 서열 13, 33, 35, 43, 59 및 61에 대해 적어도 98%의 서열 동일성을 갖는 경쇄 가변 영역을 포함하는 폴리펩티드를 코딩하는 뉴클레오티드 서열을 포함하는 핵산에 관한 것이다.
한 측면에서, 본원에 개시된 LRP6 항체는 LRP6의 프로펠러 3 영역에 결합한다. 따라서, 본 발명은 서열 69, 서열 93, 및 서열 115로 이루어진 군 중에서 선택된 CDR3을 포함하는, LRP6에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 69, 서열 93, 및 서열 115로 이루어진 군 중에서 선택된 중쇄 CDR1; 서열 70, 서열 94, 및 서열 116으로 이루어진 군 중에서 선택된 CDR2; 및 서열 71, 서열 95, 및 서열 117로 이루어진 군 중에서 선택된 CDR3을 포함하는, LRP6에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 72, 서열 96, 및 서열 118로 이루어진 군 중에서 선택된 경쇄 CDR1; 서열 73, 서열 97, 및 서열 119로 이루어진 군 중에서 선택된 CDR2; 및 서열 74, 서열 98, 및 서열 120으로 이루어진 군 중에서 선택된 CDR3을 포함하는, LRP6에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 82, 서열 89, 서열 106, 서열 108, 서열 128, 서열 130, 및 서열 138로 이루어진 군 중에서 선택된 VH; 및 서열 81, 서열 90, 서열 105, 서열 107, 서열 127, 서열 129, 및 서열 137로 이루어진 군 중에서 선택된 VL을 포함하는, LRP6에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 82, 및 서열 81을 포함하는 VL을 포함하는, LRP6에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 90을 포함하는 VH, 및 서열 89를 포함하는 VL을 포함하는, LRP6에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 106을 포함하는 VH, 및 서열 105를 포함하는 VL을 포함하는, LRP6에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 108을 포함하는 VH, 및 서열 107을 포함하는 VL을 포함하는, LRP6에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 128을 포함하는 VH, 및 서열 127을 포함하는 VL을 포함하는, LRP6에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 128을 포함하는 VH, 및 서열 127을 포함하는 VL을 포함하는, LRP6에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 130을 포함하는 VH, 및 서열 129를 포함하는 VL을 포함하는, LRP6에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 138을 포함하는 VH, 및 서열 137을 포함하는 VL을 포함하는, LRP6에 대한 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 69, 70, 71, 93, 94, 95, 115, 116, 및 117과 동일한 적어도 하나의 중쇄 CDR 서열을 포함하고 인간 LRP6 단백질에 결합하는 단리된 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 72, 73, 74, 96, 97, 98, 118, 119, 및 120과 동일한 적어도 하나의 경쇄 CDR 서열을 포함하고 인간 LRP6 단백질에 결합하는 단리된 모노클로날 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 70, 71, 93, 94, 95, 115, 116, 및 117에 대해 적어도 95%의 서열 동일성을 갖는 적어도 하나의 중쇄 CDR 서열을 포함하고 인간 LRP6 단백질에 결합하는 단리된 모노클로날 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 72, 73, 74, 96, 97, 98, 118, 119, 및 120에 대해 적어도 95%의 서열 동일성을 갖는 적어도 하나의 경쇄 CDR 서열을 포함하고 인간 LRP6 단백질에 결합하는 단리된 모노클로날 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 69의 중쇄 가변 영역 CDR1; 서열 70의 중쇄 가변 영역 CDR2; 서열 71의 중쇄 가변 영역 CDR3; 서열 72의 경쇄 가변 영역 CDR1; 서열 73의 경쇄 가변 영역 CDR2; 및 서열 74의 경쇄 가변 영역 CDR3을 포함하는 단리된 모노클로날 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 93의 중쇄 가변 영역 CDR1; 서열 94의 중쇄 가변 영역 CDR2; 서열 95의 중쇄 가변 영역 CDR3; 서열 96의 경쇄 가변 영역 CDR1; 서열 97의 경쇄 가변 영역 CDR2; 및 서열 98의 경쇄 가변 영역 CDR3을 포함하는 단리된 모노클로날 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 서열 115의 중쇄 가변 영역 CDR1; 서열 116의 중쇄 가변 영역 CDR2; 서열 117의 중쇄 가변 영역 CDR3; 서열 118의 경쇄 가변 영역 CDR1; 서열 119의 경쇄 가변 영역 CDR2; 및 서열 120의 경쇄 가변 영역 CDR3을 포함하는 단리된 모노클로날 항체 또는 그의 단편에 관한 것이고, 여기서 항체 또는 그의 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 LRP6의 β-프로펠러 1 도메인에 결합하는 모노클로날 항체 및 LRP6의 β-프로펠러 3 도메인의 조합물에 관한 것이고, 여기서 항체 또는 그의 단편은 일가이다.
한 측면에서, 본 발명은 LRP6의 β-프로펠러 1 도메인에 결합하는 모노클로날 항체 및 LRP6의 β-프로펠러 3 도메인의 조합물에 관한 것이고, 여기서 항체 또는 그의 단편은 이가이다.
한 실시양태에서, LRP6에 결합하는 항체의 단편은 Fab, F(ab2)', F(ab)2', scFv, VHH, VH, VL, dAb로 이루어지는 군 중에서 선택되고, 단편은 일가 또는 이가이다.
한 측면에서, 본 발명은 일가 또는 이가인 항체 또는 그의 단편 및 제약상 허용되는 담체를 포함하는 제약 조성물에 관한 것이다. 한 측면에서, 본 발명은 서열 82, 89, 106, 108, 128, 130, 및 138로 이루어진 군 중에서 선택된 중쇄 가변 영역을 포함하는 폴리펩티드를 코딩하는 뉴클레오티드 서열을 포함하는 핵산에 관한 것이다. 한 측면에서, 본 발명은 서열 81, 90, 105, 107, 127, 129, 및 137로 이루어진 군 중에서 선택된 경쇄 가변 영역을 포함하는 폴리펩티드를 코딩하는 뉴클레오티드 서열을 포함하는 핵산에 관한 것이다. 한 측면에서, 본 발명은 서열 82, 89, 106, 108, 128, 130, 및 138에 대해 적어도 98%의 서열 동일성을 갖는 중쇄 가변 영역을 포함하는 폴리펩티드를 코딩하는 뉴클레오티드 서열을 포함하는 핵산에 관한 것이다. 한 측면에서, 본 발명은 서열 81, 90, 105, 107, 127, 129, 및 137에 대해 적어도 98%의 서열 동일성을 갖는 경쇄 가변 영역을 포함하는 폴리펩티드를 코딩하는 뉴클레오티드 서열을 포함하는 핵산에 관한 것이다.
한 측면에서, 본 발명은 핵산을 포함하는 벡터; 및 (1) 항체의 중쇄를 코딩하는 재조합 DNA 절편 및 (2) 항체의 경쇄를 코딩하는 제2 재조합 DNA 절편을 포함하는 단리된 숙주 세포에 관한 것이다. DNA 절편은 프로모터에 작동가능하게 연결되고, 상기 숙주 세포에서 발현될 수 있다. 항체는 인간 모노클로날 항체이고; 숙주 세포는 비-인간 포유동물 세포주이다.
한 측면에서, 본 발명은 LRP6 발현 암이 있는 대상체를 선택하고, 대상체에게 유효량의 LRP6 항체 (예를 들어, 일가 또는 이가, 또는 그의 단편)를 포함하는 조성물을 투여하는 것을 포함하는 암의 치료 방법에 관한 것이다. 한 측면에서, 본 발명은 일가 또는 이가인 LRP6에 대한 항체 또는 그의 단편을 사용하여 정형적 Wnt 신호 전달 경로에 의해 매개되는 질환의 치료 방법에 관한 것이다. 한 측면에서, 본 발명은 LRP6 발현 암이 있는 대상체를 선택하고, 일가 또는 이가인 항체 또는 그의 단편을 포함하는 유효량의 조성물을 투여하는 것을 포함하는, 정형적 Wnt 신호 전달 경로에 의해 매개되는 암의 치료 방법에 관한 것이고, 여기서 암은 유방암, 폐암, 다발성 골수종, 난소암, 간암, 방광암, 위암, 전립선암, 급성 골수성 백혈병, 만성 골수성 백혈병, 골육종, 편평세포 암종, 및 흑색종으로 이루어지는 군 중에서 선택된다. 한 실시양태에서, 암은 유방암이다. 한 실시양태에서, 대상체는 인간이다.
한 측면에서, 본 발명은 LRP6 발현 암이 있는 대상체를 선택하고, 상기 대상체에게 LRP6의 프로펠러 3 영역에 결합하는 항체 또는 그의 항체 단편과 조합하여 LRP6의 프로펠러 1 영역에 결합하는 항체 또는 그의 항체 단편을 포함하는 유효량의 조성물을 투여하는 것을 포함하고, 항체 또는 그의 단편이 일가 또는 이가인 암의 치료 방법에 관한 것이다.
한 측면에서, 본 발명은 LRP6 발현 암이 있는 대상체를 선택하고, 상기 대상체에게 LRP6에 결합하여 LRP6에 의한 Wnt1 신호 전달을 억제하는 항체 또는 그의 항체 단편을 LRP6에 결합하여 LRP6에 의한 Wnt3 신호 전달을 억제하는 항체 또는 그의 항체 단편과 조합하여 포함하는 유효량의 조성물을 투여하는 것을 포함하고, 항체 또는 그의 단편이 일가 또는 이가인 암의 치료 방법에 관한 것이다.
한 측면에서, 본 발명은 숙주 세포를 배양하고, 일가 또는 이가인 항체 또는 그의 단편을 단리하는 것을 포함하는, 항체 또는 그의 단편의 생산 방법에 관한 것이다.
또 다른 측면에서, 본 발명은 유방암, 폐암, 다발성 골수종, 난소암, 간암, 방광암, 위암, 전립선암, 급성 골수성 백혈병, 만성 골수성 백혈병, 골육종, 편평세포 암종, 및 흑색종으로 이루어진 군 중에서 선택된, 정형적 Wnt 신호 전달 경로에 의해 매개되는 암의 치료를 위한 의약의 제조에 있어서 일가 또는 이가인 항체 또는 그의 단편의 용도에 관한 것이다.
한 실시양태에서, 본 발명은 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한, 서열 14의 VH 및 서열 13의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한, 서열 34의 VH 및 서열 36의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한, 서열 36의 VH 및 서열 35의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한, 서열 44의 VH 및 서열 43의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한, 서열 60의 VH 및 서열 59의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한, 서열 62의 VH 및 서열 61의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한, 서열 82의 VH 및 서열 81의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한, 서열 90의 VH 및 서열 89의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한, 서열 106의 VH 및 서열 105의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한, 서열 108의 VH 및 서열 107의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한, 서열 128의 VH 및 서열 127의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한, 서열 130의 VH 및 서열 129의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한, 서열 138의 VH 및 서열 137의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다.
한 실시양태에서, 본 발명은 약물로서 사용하기 위한, 서열 14의 VH 및 서열 13의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 약물로서 사용하기 위한, 서열 34의 VH 및 서열 33의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 약물로서 사용하기 위한, 서열 36의 VH 및 서열 35의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 약물로서 사용하기 위한, 서열 44의 VH 및 서열 43의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 약물로서 사용하기 위한, 서열 62의 VH 및 서열 61의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 약물로서 사용하기 위한, 서열 82의 VH 및 서열 81의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 약물로서 사용하기 위한, 서열 90의 VH 및 서열 89의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 약물로서 사용하기 위한, 서열 106의 VH 및 서열 105의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 약물로서 사용하기 위한, 서열 108의 VH 및 서열 107의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 약물로서 사용하기 위한, 서열 128의 VH 및 서열 127의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 약물로서 사용하기 위한, 서열 130의 VH 및 서열 129의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다. 또 다른 실시양태에서, 본 발명은 약물로서 사용하기 위한, 서열 138의 VH 및 서열 137의 VL을 갖는 일가 또는 이가인 항체에 관한 것이다.
또 다른 측면에서, 본 발명은 의약으로서 사용하기 위한 본 발명의 항체 또는 그의 단편에 관한 것이다. 또 다른 측면에서, 본 발명은 LRP6 발현 암의 치료를 위한 의약으로서 사용하기 위한 본 발명의 항체 또는 그의 단편에 관한 것이다. 한 측면에서, 본 발명은 유방암, 폐암, 다발성 골수종, 난소암, 간암, 방광암, 위암, 전립선암, 급성 골수성 백혈병, 만성 골수성 백혈병, 골육종, 편평세포 암종, 및 흑색종으로 이루어진 군 중에서 선택된 LRP6 발현 암의 치료를 위한 의약으로서 사용하기 위한 본 발명의 항체 또는 그의 단편에 관한 것이다.
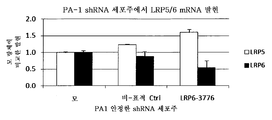
도 1은 PA1 세포, U266 세포 및 다우디 (Daudi) 세포에 대한 선택된 Fab의 FACS EC50 결정 및 상응하는 mRNA 발현 데이터 (aa 및 ab) 및 shRNA에 의한 LRP6의 녹다운 (knockdown) 및 상응하는 mRNA 발현 데이터 (ba 및 bb)를 보여주는 그래프이다;
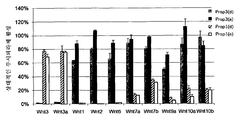
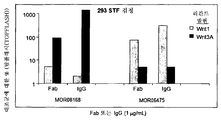
도 2a-b는 Wnt1 또는 Wnt3A 리간드를 발현하는 HEK293T/17 STF 세포 (유전자 리포터 검정)에서 항-LRP6 Fab 단편 활성을 보여주는 그래프이다. 데이터는 항-LRP6 Fab가 Wnt1 또는 Wnt3 신호 전달을 선택적으로 차단함을 보여준다;
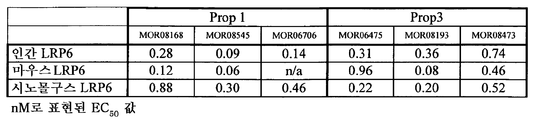
도 3은 인간, 마우스 및 시노몰구스 원숭이에 대한 항-LRP6 β-프로펠러 1 및 β-프로펠러 3 항체의 교차-반응성 값을 보여준다;
도 4는 HEK293T/17 STF 세포에서 다양한 WNT 리간드의 일시적 발현 (유전자 리포터 검정) 및 항-LRP6 항체로의 처리를 보여주는 그래프로서, LRP6의 특이적 β-프로펠러 영역에 대한 항체 결합/차단을 기초로 한 특정 WNT의 활성 억제를 보여준다;
도 5는 IgG로의 Fab 전환이 비-차단된 Wnt 리간드로부터 신호의 강화를 야기함을 보여주는 막대 그래프이다;
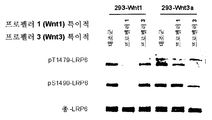
도 6은 세포 시스템에서 LRP6의 선택적인 표적 억제를 보여주는 웨스턴 블롯 (western blot)이다;
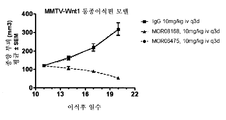
도 7은 설치류에서 5 mg/kg에서 β-프로펠러 1 영역에 결합하는 LRP6 항체의 단일 i.v. 용량을 보여주는 그래프이다;
도 8aa-8ac는 <0.01의 조정된 P-값에서 t=0 대조군에 비해 >2배 상향조절된 MMTV-Wnt1 종양 내의 유전자를 보여주는 표이고, 도 8ba-8be는 단일 용량의 MOR08168 (5 mg/kg)을 MMTV-Wnt1 종양 보유 마우스에게 투여하고 8 h 후에 <0.01의 조정된 P-값에서 t=0 대조군에 비해 >2배 하향조절된 유전자를 보여주는 표이다;
도 9a는 프로펠러 3 mAb가 아니라 프로펠러 1 mAb가 MMTV-Wnt1 모델에서 생체내 종양 퇴행을 야기함을 보여주는 그래프이다. 도 9b는 MMTV-Wnt1 종양 모델의 성장에 대한 상이한 용량의 프로펠러 1 mAb의 효과를 보여주는 그래프이다;
도 10은 프로펠러 1 mAb가 아니라 프로펠러 3 mAb가 MMTV-Wnt3 모델에서 종양 성장의 억제를 야기함을 보여주는 그래프이다.
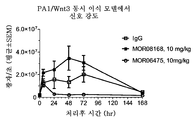
도 11은 프로펠러 1 mAb가 아니라 프로펠러 3 mAb가 생체 내에서 PA-1 세포에서 Wnt3A-유도 수퍼 탑 플래시 (Super Top Flash) 활성의 억제를 야기함을 보여주는 그래프이다.
도 12는 HDx MS에 의한 MOR06475에 의한 LRP6 PD3-4의 용매 보호된 영역을 보여주는 도면이고 (a), 특이적 잔기의 돌연변이가 scFv MOR06475의 결합 상실을 야기함을 보여준다 (b);
도 13은 LRP6의 β-프로펠러 영역을 보여주는 모식도이다;
도 14는 모든 scFv 분자가 이. 콜라이 (E. coli)로부터 성공적으로 발현되고 정제됨을 보여주는 SDS-PAGE 겔의 사진이다;
도 15A-D는 다가 항체의 모식적인 예이다. (15A) 완전 IgG의 C-말단에 부착된 scFv scFv, (15B) Fc의 N-말단에 부착된 scFv scFv, (15C) Fc의 C-말단에 부착된 scFv scFv, (15D) Fc의 N 및 C 말단에 부착된 scFv scFv;
도 16은 비-환원 (레인 1) 및 환원 (레인 2) 조건 하에 정제된 이중파라토프 항-LRP6 IgG scFv를 보여주는 SDS-PAGE 겔의 사진이다;
도 17은 이중파라토프 항체 및 별개의 각각의 성분 부분의 STF 검정에서의 활성을 보여준다;
도 18은 scFv 분자에서 링커 길이 비교의 STF 검정에서의 활성을 보여준다;
도 19는 이중파라토프 항체의 결합 활성을 보여주는 표이다;
도 20은 PA-1/Wnt3a L-세포 동시 배양 모델에서 이중파라토프 항체 및 Prop3 항체 (Prop1 항체가 아니라)의 활성을 보여준다;
도 21은 설치류에서 5 mg/kg의 Prop1 LRP6 항체 및 Prop1/3 이중파라토프 항체의 단일 i.v. 용량 사이의 비교를 보여주는 그래프이다;
도 22는 프로펠러 1 및 이중파라토프 프로펠러 1/3 항체 둘 모두가 MMTV-Wnt1 모델에서 생체내 종양 퇴행을 야기함을 보여주는 그래프이다;
도 23은 MMTV-Wnt1 모델에서 Prop1/3 결합 이중파라토프 항체의 용량-반응 관계를 보여주는 그래프이다;
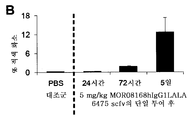
도 24는 뮤린 MMTV-Wnt1 유방 종양의 분화가 길항작용성 LRP6 항체에 의해 유도됨을 보여준다. a-b) MMTV-Wnt1 종양의 단편을 누드 (nude) 마우스 내로 피하 이식하였다. 종양 함유 마우스를 단일 용량의 PBS (대조군) 또는 5 mg/kg MOR08168IgG1LALA 6475 scfv로 처리하였다. a) 지질에 대한 오일 레드 (Oil Red) O 염색의 대표적인 영상. b) 오일 레드 O 염색의 정량. 그래프는 평균±SEM 값을 나타낸다. 72시간 군에서 n=4, 24시간 군에서 n=3, 5일 군에서 n=2, 및 PBS (대조군)에 대해 n=1;
도 25는 E-카드헤린 (Cadherin) 음성 MDA-MB231 이종이식편 모델에서 Prop1/3 결합 이중파라토프 항체의 활성을 보여주는 그래프이다;
도 26은 재조합 LRP6 PD1/2 및 PD3/4에 대한 MOR08168, MOR06475 및 MOR08168IgG1LALA 6475 scfv의 친화도 및 결합 동역학을 보여준다. a) 비아코어 (Biacore) 분석에 의해 결정한 친화도 및 온/오프 (on/off) 속도의 요약 표. ba-bb) 상응하는 LRP6 수용체 도메인, PD1/2 및 PD3/4에 대한 항-LRP6 분자의 대표적인 결합 곡선. c) LRP6 PD1/2 및 PD3/4의 MOR08168IgG1LALA 6475 scfv에 대한 순차적인 결합;
도 27은 IgG 기반 이중파라토프 항체의 모식도를 보여준다;
도 28은 이. 콜라이에서 항-LRP6 scFv 발현의 최적화를 보여주는 SDS-PAGE 겔의 사진이다;
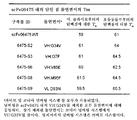
도 29는 Tm에 대한 MOR06475 scFv의 단일 돌연변이의 효과를 보여주는 표이다;
도 30은 Tm에 대한 MOR08168 scFv의 단일 돌연변이의 효과를 보여주는 표이다.
도 31은 박테리아와 포유동물 시스템 모두에서 발현되는 물질의 Tm에 대한 MOR08168 scFv의 이중 돌연변이의 효과를 보여주는 표이다;
도 32는 ELISA, 프로테온 (Proteon) 친화도 및 STF 리포터 유전자 검정에서 MOR06475 및 MOR08168 scFv의 야생형 및 단일/이중 돌연변이된 버전의 결합 및 기능적 활성을 요약하는 표이다;
도 33은 고안된 돌연변이의 선택된 예의 제시한다. 모든 도면에서, 단백질 백본은 리본 그림으로 표현되고, 선택된 측쇄는 막대로 표현된다. (a): scFv6475의 상동성 모델에서, VH:I37은 VL:F98 및 VH:W103인 2개의 방향족 잔기에 근접한다. (b) scFv6475의 VH:I37F 돌연변이체에서, VH:F37 및 VH:W103은 수직의 파이-파이 적층 (pi-pi stacking) 상호작용을 형성할 수 있었고, VH:F37 및 VL:F98은 또 다른 수직의 파이-파이 적층 상호작용을 형성할 수 있었다. (c) scFv8168의 상동성 모델에서, 소수성 잔기 VH:V33은 극성 잔기 VH:N100a에 근접한다. (d) scFv8168의 VH:V33N 돌연변이체에서, VH:N33 측쇄는 상동성 모델링에서 제시되는, VH:N100a와 수소 결합을 형성할 수 있다. 2개의 잔기들 사이의 수소 결합은 결합에 의해 제시된다. (e): scFv8168의 상동성 모델에서, 하전된 잔기 VH:K43은 소수성 잔기 VH:V85와 염 가교 (salt bridge)를 형성하지 않았다. (f): VH:K43 및 VH:E85의 2개의 하전된 측쇄는 scFv8168에 대한 VH:V85E 돌연변이 때문에 염 가교를 형성할 수 있다. 2개의 하전 기 사이의 거리는 2.61 Å일 수 있다;
도 34는 이중파라토프 항체의 열안정성 측정치를 보여주는 표이다.
도 2a-b는 Wnt1 또는 Wnt3A 리간드를 발현하는 HEK293T/17 STF 세포 (유전자 리포터 검정)에서 항-LRP6 Fab 단편 활성을 보여주는 그래프이다. 데이터는 항-LRP6 Fab가 Wnt1 또는 Wnt3 신호 전달을 선택적으로 차단함을 보여준다;
도 3은 인간, 마우스 및 시노몰구스 원숭이에 대한 항-LRP6 β-프로펠러 1 및 β-프로펠러 3 항체의 교차-반응성 값을 보여준다;
도 4는 HEK293T/17 STF 세포에서 다양한 WNT 리간드의 일시적 발현 (유전자 리포터 검정) 및 항-LRP6 항체로의 처리를 보여주는 그래프로서, LRP6의 특이적 β-프로펠러 영역에 대한 항체 결합/차단을 기초로 한 특정 WNT의 활성 억제를 보여준다;
도 5는 IgG로의 Fab 전환이 비-차단된 Wnt 리간드로부터 신호의 강화를 야기함을 보여주는 막대 그래프이다;
도 6은 세포 시스템에서 LRP6의 선택적인 표적 억제를 보여주는 웨스턴 블롯 (western blot)이다;
도 7은 설치류에서 5 mg/kg에서 β-프로펠러 1 영역에 결합하는 LRP6 항체의 단일 i.v. 용량을 보여주는 그래프이다;
도 8aa-8ac는 <0.01의 조정된 P-값에서 t=0 대조군에 비해 >2배 상향조절된 MMTV-Wnt1 종양 내의 유전자를 보여주는 표이고, 도 8ba-8be는 단일 용량의 MOR08168 (5 mg/kg)을 MMTV-Wnt1 종양 보유 마우스에게 투여하고 8 h 후에 <0.01의 조정된 P-값에서 t=0 대조군에 비해 >2배 하향조절된 유전자를 보여주는 표이다;
도 9a는 프로펠러 3 mAb가 아니라 프로펠러 1 mAb가 MMTV-Wnt1 모델에서 생체내 종양 퇴행을 야기함을 보여주는 그래프이다. 도 9b는 MMTV-Wnt1 종양 모델의 성장에 대한 상이한 용량의 프로펠러 1 mAb의 효과를 보여주는 그래프이다;
도 10은 프로펠러 1 mAb가 아니라 프로펠러 3 mAb가 MMTV-Wnt3 모델에서 종양 성장의 억제를 야기함을 보여주는 그래프이다.
도 11은 프로펠러 1 mAb가 아니라 프로펠러 3 mAb가 생체 내에서 PA-1 세포에서 Wnt3A-유도 수퍼 탑 플래시 (Super Top Flash) 활성의 억제를 야기함을 보여주는 그래프이다.
도 12는 HDx MS에 의한 MOR06475에 의한 LRP6 PD3-4의 용매 보호된 영역을 보여주는 도면이고 (a), 특이적 잔기의 돌연변이가 scFv MOR06475의 결합 상실을 야기함을 보여준다 (b);
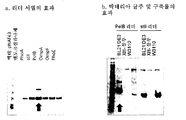
도 13은 LRP6의 β-프로펠러 영역을 보여주는 모식도이다;
도 14는 모든 scFv 분자가 이. 콜라이 (E. coli)로부터 성공적으로 발현되고 정제됨을 보여주는 SDS-PAGE 겔의 사진이다;
도 15A-D는 다가 항체의 모식적인 예이다. (15A) 완전 IgG의 C-말단에 부착된 scFv scFv, (15B) Fc의 N-말단에 부착된 scFv scFv, (15C) Fc의 C-말단에 부착된 scFv scFv, (15D) Fc의 N 및 C 말단에 부착된 scFv scFv;
도 16은 비-환원 (레인 1) 및 환원 (레인 2) 조건 하에 정제된 이중파라토프 항-LRP6 IgG scFv를 보여주는 SDS-PAGE 겔의 사진이다;
도 17은 이중파라토프 항체 및 별개의 각각의 성분 부분의 STF 검정에서의 활성을 보여준다;
도 18은 scFv 분자에서 링커 길이 비교의 STF 검정에서의 활성을 보여준다;
도 19는 이중파라토프 항체의 결합 활성을 보여주는 표이다;
도 20은 PA-1/Wnt3a L-세포 동시 배양 모델에서 이중파라토프 항체 및 Prop3 항체 (Prop1 항체가 아니라)의 활성을 보여준다;
도 21은 설치류에서 5 mg/kg의 Prop1 LRP6 항체 및 Prop1/3 이중파라토프 항체의 단일 i.v. 용량 사이의 비교를 보여주는 그래프이다;
도 22는 프로펠러 1 및 이중파라토프 프로펠러 1/3 항체 둘 모두가 MMTV-Wnt1 모델에서 생체내 종양 퇴행을 야기함을 보여주는 그래프이다;
도 23은 MMTV-Wnt1 모델에서 Prop1/3 결합 이중파라토프 항체의 용량-반응 관계를 보여주는 그래프이다;
도 24는 뮤린 MMTV-Wnt1 유방 종양의 분화가 길항작용성 LRP6 항체에 의해 유도됨을 보여준다. a-b) MMTV-Wnt1 종양의 단편을 누드 (nude) 마우스 내로 피하 이식하였다. 종양 함유 마우스를 단일 용량의 PBS (대조군) 또는 5 mg/kg MOR08168IgG1LALA 6475 scfv로 처리하였다. a) 지질에 대한 오일 레드 (Oil Red) O 염색의 대표적인 영상. b) 오일 레드 O 염색의 정량. 그래프는 평균±SEM 값을 나타낸다. 72시간 군에서 n=4, 24시간 군에서 n=3, 5일 군에서 n=2, 및 PBS (대조군)에 대해 n=1;
도 25는 E-카드헤린 (Cadherin) 음성 MDA-MB231 이종이식편 모델에서 Prop1/3 결합 이중파라토프 항체의 활성을 보여주는 그래프이다;
도 26은 재조합 LRP6 PD1/2 및 PD3/4에 대한 MOR08168, MOR06475 및 MOR08168IgG1LALA 6475 scfv의 친화도 및 결합 동역학을 보여준다. a) 비아코어 (Biacore) 분석에 의해 결정한 친화도 및 온/오프 (on/off) 속도의 요약 표. ba-bb) 상응하는 LRP6 수용체 도메인, PD1/2 및 PD3/4에 대한 항-LRP6 분자의 대표적인 결합 곡선. c) LRP6 PD1/2 및 PD3/4의 MOR08168IgG1LALA 6475 scfv에 대한 순차적인 결합;
도 27은 IgG 기반 이중파라토프 항체의 모식도를 보여준다;
도 28은 이. 콜라이에서 항-LRP6 scFv 발현의 최적화를 보여주는 SDS-PAGE 겔의 사진이다;
도 29는 Tm에 대한 MOR06475 scFv의 단일 돌연변이의 효과를 보여주는 표이다;
도 30은 Tm에 대한 MOR08168 scFv의 단일 돌연변이의 효과를 보여주는 표이다.
도 31은 박테리아와 포유동물 시스템 모두에서 발현되는 물질의 Tm에 대한 MOR08168 scFv의 이중 돌연변이의 효과를 보여주는 표이다;
도 32는 ELISA, 프로테온 (Proteon) 친화도 및 STF 리포터 유전자 검정에서 MOR06475 및 MOR08168 scFv의 야생형 및 단일/이중 돌연변이된 버전의 결합 및 기능적 활성을 요약하는 표이다;
도 33은 고안된 돌연변이의 선택된 예의 제시한다. 모든 도면에서, 단백질 백본은 리본 그림으로 표현되고, 선택된 측쇄는 막대로 표현된다. (a): scFv6475의 상동성 모델에서, VH:I37은 VL:F98 및 VH:W103인 2개의 방향족 잔기에 근접한다. (b) scFv6475의 VH:I37F 돌연변이체에서, VH:F37 및 VH:W103은 수직의 파이-파이 적층 (pi-pi stacking) 상호작용을 형성할 수 있었고, VH:F37 및 VL:F98은 또 다른 수직의 파이-파이 적층 상호작용을 형성할 수 있었다. (c) scFv8168의 상동성 모델에서, 소수성 잔기 VH:V33은 극성 잔기 VH:N100a에 근접한다. (d) scFv8168의 VH:V33N 돌연변이체에서, VH:N33 측쇄는 상동성 모델링에서 제시되는, VH:N100a와 수소 결합을 형성할 수 있다. 2개의 잔기들 사이의 수소 결합은 결합에 의해 제시된다. (e): scFv8168의 상동성 모델에서, 하전된 잔기 VH:K43은 소수성 잔기 VH:V85와 염 가교 (salt bridge)를 형성하지 않았다. (f): VH:K43 및 VH:E85의 2개의 하전된 측쇄는 scFv8168에 대한 VH:V85E 돌연변이 때문에 염 가교를 형성할 수 있다. 2개의 하전 기 사이의 거리는 2.61 Å일 수 있다;
도 34는 이중파라토프 항체의 열안정성 측정치를 보여주는 표이다.
본 발명의 상세한 설명
정의
본 발명을 보다 쉽게 이해할 수 있도록, 특정 용어를 먼저 규정한다. 추가의 정의는 상세한 설명 전체에 걸쳐 제시한다.
본원에서 사용되는 구문 "면역 반응"은 인체로부터 침습 병원체, 병원체로 감염된 세포 또는 조직, 암성 세포, 또는, 자가면역 또는 병리학적 염증의 경우, 정상 인간 세포 또는 조직의 선택적인 손상, 파괴, 또는 제거를 유도하는, 예를 들어 림프구, 항원 제시 세포, 포식 세포, 과립구, 및 상기 세포 또는 간 (항체, 시토카인, 및 보체 포함)에 의해 생산되는 가용성 거대분자의 작용을 의미한다.
본원에서 사용되는 구문 "신호 전달 경로" 또는 "신호 전달 활성"은 세포의 한 부분으로부터 세포의 또 다른 부분으로 신호의 전달을 유발하는 단백질-단백질 상호작용, 예컨대 성장 인자의 수용체에 대한 결합에 의해 일반적으로 개시되는 생화학적 인과 관계를 의미한다. LRP6에 대해, 전달은 신호 전달을 야기하는 일련의 반응에서 하나 이상의 단백질 상의 하나 이상의 티로신, 세린, 또는 트레오닌 잔기의 특이적 인산화를 수반한다. 끝에서 두 번째의 과정은 일반적으로 유전자 발현의 변경을 야기하는 핵 사건을 포함한다.
본원에서 사용되는 구문 "Wnt 신호 전달 경로"는 분비된 단백질 리간드의 Wnt 패밀리의 멤버가 β-카테닌의 핵 내로의 전위를 허용하는 LRP 및 프리즐드 (FZD)의 수용체 복합체에 결합하고, LEF/TCF 전사 인자와 상호작용하고, 표적 유전자 발현을 활성화하는 정형적 Wnt 경로를 의미한다. Wnt 신호 전달 경로는 Wnt 리포터 유전자 검정 또는 본원에서 설명되는 Wnt 지정 신호 전달의 다른 척도 (예를 들어, LRP6 인산화, β-카테닌 안정화 및 핵 전위, 세포 증식/생존)를 사용하여 측정할 수 있다.
구문 "Wnt1 신호 전달 경로"는 Wnt1 리간드 및 Wnt1 결합 리간드의 클래스, 예컨대 Wnt2, Wnt6, Wnt7a, Wnt7b, Wnt9a, Wnt10a, 또는 Wnt10b와 상호작용하는 LRP6에 의해 활성화되는 정형적 Wnt 경로를 의미한다.
구문 "Wnt3 신호 전달 경로"는 Wnt3 또는 Wnt3a 리간드와 상호작용하는 LRP6에 의해 활성화되는 정형적 Wnt 경로를 의미한다.
용어 "LRP6"은 기탁 번호 NP002327에서 규정되는 인간 LRP6을 의미한다.
본원에서 사용되는 용어 "항체"는 LRP6 에피토프와 상호작용하고 (예를 들어, 결합, 입체 장애, 안정화/탈안정화, 공간 분포에 의해) 신호 전달을 억제하는 전체 항체를 의미한다. 천연 생성 "항체"는 디술피드 결합에 의해 서로 연결된 적어도 2개의 중쇄 (H) 및 2개의 경쇄 (L)를 포함하는 당단백질이다. 각각의 중쇄는 중쇄 가변 영역 (본원에서 VH로서 약칭함) 및 중쇄 불변 영역으로 이루어진다. 중쇄 불변 영역은 3개의 도메인 CH1, CH2 및 CH3으로 이루어진다. 각각의 경쇄는 경쇄 가변 영역 (본원에서 VL로 약칭함) 및 경쇄 불변 영역으로 이루어진다. 경쇄 불변 영역은 하나의 도메인 CL로 이루어진다. VH 및 VL 영역은 프레임워크 영역 (FR)으로 불리는 보다 보존된 영역이 산재하는, 상보성 결정 영역 (CDR)으로 불리는 초가변성 영역으로 추가로 하위분류될 수 있다. 각각의 VH 및 VL은 아미노-말단으로부터 카르복시-말단으로 다음 순서로 배열된 3개의 CDR 및 4개의 FR로 이루어진다: FR1, CDR1, FR2, CDR2, FR3, CDR3, FR4. 중쇄 및 경쇄의 가변 영역은 항원과 상호작용하는 결합 도메인을 함유한다. 항체의 불변 영역은 면역계의 다양한 세포 (예를 들어, 이펙터 세포) 및 전통적인 보체계의 제1 성분 (C1q)을 비롯한 숙주 조직 또는 인자에 대한 이뮤노글로불린의 결합을 매개할 수 있다. 용어 "항체"는 예를 들어 모노클로날 항체, 인간 항체, 인간화 항체, 낙타화 (camelised) 항체, 키메라 항체, 단일 쇄 Fv (scFv), 디술피드-연결 Fv (sdFv), Fab 단편, F(ab') 단편, 및 항-이디오타입 (항-Id) 항체 (예를 들어, 본 발명의 항체에 대한 항-Id 항체 포함), 및 임의의 상기 항체의 에피토프-결합 단편을 포함한다. 항체는 임의의 이소형 (예를 들어, IgG, IgE, IgM, IgD, IgA 및 IgY), 클래스 (예를 들어, IgG1, IgG2, IgG3, IgG4, IgA1 및 IgA2) 또는 서브클래스의 것일 수 있다.
경쇄 및 중쇄는 모두 구조적 및 기능적 상동성 영역으로 나눌 수 있다. 용어 "불변" 및 "가변"은 기능적으로 사용된다. 이와 관련하여, 경쇄 (VL) 및 중쇄 (VH) 부분 둘 모두의 가변 도메인이 항원 인식 및 특이성을 결정함이 이해될 것이다. 이와 반대로, 경쇄의 불변 도메인 (CL) 및 중쇄의 불변 도메인 (CH1, CH2 또는 CH3)은 중요한 생물학적 특성, 예컨대 분비, 태반 경유 이동성, Fc 수용체 결합, 보체 결합 등을 부여한다. 관례상, 불변 영역 도메인의 넘버링은 이들이 항체의 항원 결합 부위 또는 아미노-말단으로부터 보다 멀어지면서 증가한다. N-말단은 가변 영역이고 C-말단은 불변 영역이고; CH3 및 CL 도메인은 실제로 각각 중쇄 및 경쇄의 카르복시-말단을 포함한다.
본원에서 사용되는 구문 "항체 단편"은 LRP6 에피토프와 (예를 들어, 결합, 입체 장애, 안정화/탈안정화, 공간 분포에 의해) 특이적으로 상호작용하고 신호 전달을 억제하는 능력을 보유하는 항체의 하나 이상의 부분을 의미한다. 결합 단편의 예는 Fab 단편, VL, VH, CL 및 CH1 도메인으로 이루어지는 일가 단편; F(ab)2 단편, 즉 힌지 영역에서 디술피드 다리에 의해 연결된 2개의 Fab 단편을 포함하는 이가 단편; VH 및 CH1 도메인으로 이루어지는 Fd 단편; 항체의 단일 아암 (arm)의 VL 및 VH 도메인으로 이루어지는 Fv 단편; VH 도메인으로 이루어지는 dAb 단편 (Ward et al., (1989) Nature 341:544-546); 및 단리된 상보성 결정 영역 (CDR)을 포함하고 이로 제한되지 않는다.
또한, Fv 단편의 2개의 도메인 VL 및 VH가 별개의 유전자에 의해 코딩되지만, 이들은 재조합 방법을 이용하여, VL 및 VH 영역이 페어링되어 일가 분자를 형성하는 단일 단백질 사슬 (단일 쇄 Fv (scFv)로 알려짐; 예를 들어, 문헌 [Bird et al., (1988) Science 242:423-426]; 및 [Huston et al., (1988) Proc. Natl. Acad. Sci. 85:5879-5883] 참조)로 제조될 수 있도록 하는 합성 링커에 의해 연결될 수 있다. 또한, 상기 단일 쇄 항체는 용어 "항체 단편"에 포함되는 것으로 의도된다. 상기 항체 단편은 당업자에게 공지된 통상적인 기술을 사용하여 얻고, 단편은 무손상 항체와 동일한 방식으로 유용성에 대해 스크리닝된다.
항체 단편은 또한 부분은 또한 단일 도메인 항체, 맥시바디 (maxibody), 미니바디 (minibody), 디아바디, 트리아바디 (triabody), 테트라바디 (tetrabody), v-NAR 및 비스-scFv 내로 혼입될 수도 있다 (예를 들어, 문헌 [Hollinger and Hudson, (2005), Nature Biotechnology, 23: 1126-1136] 참조). 항체 단편은 피브로넥틴 유형 III (Fn3)과 같은 폴리펩티드를 기초로 하는 스캐폴드 내로 그라프팅될 수 있다 (피브로넥틴 폴리펩티드 모노바디 (monobody)를 기재하고 있는 미국 특허 번호 6,703,199 참조).
항체 단편은 상보성 경쇄 폴리펩티드와 함께 항원 결합 영역 쌍을 형성하는 직렬식 Fv 절편 (VH-CH1-VH-CH1)의 쌍을 포함하는 단일 쇄 분자 내로 혼입될 수 있다 ([Zapata et al., (1995) Protein Eng. 8:1057-1062]; 및 미국 특허 번호 5,641,870).
용어 "다가 항체"는 1 초과의 결합가를 갖는 단일 결합 분자를 의미하고, 여기서, "결합가"는 항체 구축물의 분자당 존재하는 항원-결합 모이어티 (moiety)의 수로서 설명된다. 따라서, 단일 결합 분자는 표적 수용체 상의 1 초과의 결합 부위에 결합할 수 있다. 다가 항체의 예는 이가 항체, 3가 항체, 4가 항체, 5가 항체 등, 및 이중특이적 항체 및 이중파라토프 항체를 포함하고 이로 제한되지 않는다. 예를 들어, LRP6 수용체에 대해, 다가 항체 (예를 들어, LRP6 이중파라토프 항체)는 각각 LRP6의 β-프로펠러 1 도메인 결합 부위에 대한 결합 모이어티 및 β-프로펠러 3 도메인 결합 부위의 결합 모이어티를 갖는다.
또한, 용어 "다가 항체"는 2개의 별개의 표적 수용체에 대한 하나 초과의 항원-결합 모이어티를 갖는 단일 결합 분자, 예를 들어, LRP6 표적 수용체 및 LRP6이 아닌 제2 표적 수용체 (예컨대 ErbB, cmet, IGFR1, 스무든드 (Smoothened), 노치 (Notch) 수용체) 둘 모두에 결합하는 항체를 의미한다. 하나의 실시양태에서, 다가 항체는 4개의 수용체 결합 도메인을 갖는 4가 항체이다. 4가 분자는 표적 수용체 상의 각각의 결합 부위에 대해 이중특이적 및 이가일 수 있다.
다가 항체는 생물학적 효과를 매개하고, 예를 들어, 세포 활성화를 조정하여 (예를 들어, 세포 표면 수용체에 결합하고 활성화 또는 억제성 신호의 전달 또는 억제를 유발함으로써), 세포 사멸을 야기하거나 (예를 들어, 세포 신호 유도 경로에 의해), 또는 대상체에서 질환 또는 장애를 조절한다 (예를 들어, 세포 사멸을 매개 또는 촉진함으로써, 또는 생체이용가능한 물질의 양을 조정함으로써).
본원에서 사용되는 용어 "일가 항체"는 표적 수용체, 예컨대 LRP6 상의 단일 에피토프에 결합하는 항체를 의미한다.
본원에서 사용되는 용어 "이가 항체"는 적어도 2개의 동일한 표적 수용체 상의 2개의 에피토프에 결합하는 항체 (예를 들어, 2개의 LRP6 수용체의 β-프로펠러 1 도메인에 결합하는 항체, 또는 2개의 LRP6 수용체의 β-프로펠러 3 도메인에 결합하는 항체)를 의미한다. 또한, 이가 항체는 표적 수용체를 서로 가교결합시킬 수 있다. "이가 항체"는 또한 적어도 2개의 동일한 표적 수용체 상의 2개의 상이한 에피토프에 결합하는 항체를 의미한다.
본원에서 사용되는 용어 "이중파라토프 항체"는 동일한 표적 수용체 상의 2개의 상이한 에피토프에 결합하는 항체, 예를 들어, 단일 LRP6 수용체의 β-프로펠러 1 도메인 및 β-프로펠러 3 도메인에 결합하는 항체를 의미한다. 또한, 이 용어는 적어도 2개의 LRP6 수용체(들)의 β-프로펠러 1 및 β-프로펠러 3 도메인 둘 모두에 결합하는 항체를 포함한다.
본원에서 사용되는 용어 "이중특이적 항체"는 적어도 2개의 상이한 표적 수용체 (예를 들어, LRP6 수용체 및 LRP6 수용체가 아닌 수용체) 상의 2개 이상의 상이한 에피토프에 결합하는 항체를 의미한다.
본원에서 사용되는 구문 "단리된 항체"는 상이한 항원 특이성을 갖는 다른 항체가 실질적으로 없는 항체를 의미한다 (예를 들어, LRP6에 특이적으로 결합하는 단리된 항체에는 LRP6 이외의 항원에 특이적으로 결합하는 항체가 실질적으로 없음). 또한, 단리된 항체에는 다른 세포성 물질 및/또는 화학물질이 실질적으로 없을 수 있다. 그러나, LRP6에 특이적으로 결합하는 단리된 항체는 다른 항원, 예컨대 다른 종으로부터의 LRP6 분자에 대한 교차 반응성을 가질 수 있다. 추가로, 단리된 항체에는 다른 세포 물질 및/또는 화학물질이 실질적으로 없을 수 있다.
본원에서 사용되는 구문 "모노클로날 항체" 또는 "모노클로날 항체 조성물"은 실질적으로 동일한 아미노산 서열을 갖거나 또는 동일한 유전자 공급원으로부터 유래된 항체, 항체 단편, 이중특이적 항체 등을 포함하는 폴리펩티드를 의미한다. 또한, 상기 용어는 단일 분자 조성물의 항체 분자의 제제를 의미한다. 모노클로날 항체 조성물은 특정 에피토프에 대한 단일 결합 특이성 및 친화도를 보인다.
본원에서 사용되는 구문 "인간 항체"는 프레임워크 영역과 CDR 영역 둘 모두가 인간 기원의 서열로부터 유래된 가변 영역을 갖는 항체를 포함한다. 추가로, 항체가 불변 영역을 함유하는 경우, 상기 불변 영역 역시 이러한 인간 서열, 예를 들어 인간 배선 서열, 또는 인간 배선 서열의 돌연변이된 버전 또는 예를 들어 문헌 [Knappik, et al. (2000. J Mol Biol 296, 57-86)]에 기재된 바와 같이 인간 프레임워크 서열 분석으로부터 유래된 컨센서스 (consensus) 프레임워크 서열을 함유하는 항체로부터 유래된다. 이뮤노글로불린 가변 도메인, 예를 들어, CDR의 구조 및 위치는 공지된 넘버링 방식, 예를 들어, 카바트 (Kabat) 넘버링 방식, 코티아 (Chothia) 넘버링 방식, 또는 카바트 및 코티아의 조합을 사용하여 규정될 수 있다 (예를 들어, 문헌 [Sequences of Proteins of Immunological Interest, U.S. Department of Health and Human Services (1991), eds. Kabat et al.]; [Al Lazikani et al., (1997) J. Mol. Bio. 273:927 948]; [Kabat et al., (1991) Sequences of Proteins of Immunological Interest, 5th edit., NIH Publication no. 91-3242 U.S. Department of Health and Human Services]; [Chothia et al., (1987) J. Mol. Biol. 196:901-917]; [Chothia et al., (1989) Nature 342:877-883]; 및 [Al-Lazikani et al., (1997) J. Mol. Biol. 273:927-948] 참조).
본 발명의 인간 항체는 인간 서열에 의해 코딩되지 않는 아미노산 잔기 (예를 들어, 시험관내 무작위 또는 부위-특이적 돌연변이 유발 또는 생체내 체세포 돌연변이에 의해 도입된 돌연변이, 또는 안정성 또는 제조를 용이하게 하기 위한 보존적 치환)를 포함할 수 있다. 그러나, 본원에서 사용되는 용어 "인간 항체"는 또 다른 포유동물 종, 예컨대 마우스의 배선으로부터 유래된 CDR 서열이 인간 프레임워크 서열 상에 그라프팅된 항체를 포함하고자 의도되지 않는다.
본원에서 사용되는 구문 "인간 모노클로날 항체"는 프레임워크 및 CDR 영역 둘 모두가 인간 서열로부터 유래된 가변 영역을 갖는 단일 결합 특이성을 보이는 항체를 의미한다. 한 실시양태에서, 인간 모노클로날 항체는 트랜스제닉 비인간 동물, 예를 들어, 불멸화 세포에 융합된 인간 중쇄 트랜스진 및 경쇄 트랜스진를 포함하는 게놈을 갖는 트랜스제닉 마우스로부터 얻은 B 세포를 포함하는 하이브리도마에 의해 생산된다.
본원에서 사용되는 구문 "재조합 인간 항체"는 재조합 수단에 의해 제조, 발현, 생성 또는 단리된 모든 인간 항체, 예컨대 인간 이뮤노글로불린 유전자에 대한 트랜스제닉 (transgenic) 또는 트랜스크로모조멀 (transchromosomal)인 동물 (예를 들어 마우스), 또는 이로부터 제조된 하이브리도마로부터 단리된 항체, 인간 항체를 발현하도록 형질전환된 숙주 세포, 예를 들어 트랜스펙토마 (transfectoma)로부터 단리된 항체, 재조합의 조합형 인간 항체 라이브러리로부터 단리된 항체, 및 인간 이뮤노글로불린 유전자 서열의 전부 또는 일부를 다른 DNA 서열로 스플라이싱하는 것을 포함하는 임의의 다른 수단에 의해 제조, 발현, 생성 또는 단리된 항체를 포함한다. 이러한 재조합 인간 항체는 프레임워크 및 CDR 영역이 인간 배선 이뮤노글로불린 서열로부터 유래된 가변 영역을 갖는다. 그러나, 특정 실시양태에서, 이러한 재조합 인간 항체를 대상으로 하여 시험관내 돌연변이 유발 (또는, 인간 Ig 서열에 대해 트랜스제닉인 동물을 사용하는 경우에는 생체내 체세포 돌연변이 유발)을 행할 수 있고, 따라서 재조합 항체의 VH 및 VL 영역의 아미노산 서열은, 인간 배선 VH 및 VL 서열로부터 유래되고 이와 관련이 있지만 생체 내에서 인간 항체 배선 레퍼토리 내에 천연적으로 존재하지 않을 수 있는 서열이다.
본원에서 사용되는 용어 "링커"는 scFv를 IgG에 연결하기 위해 사용되는 글리신 및 세린 잔기로 이루어지는 펩티드 링커를 의미한다. 예시적인 Gly/Ser 링커는 아미노산 서열 (Gly-Gly-Ser)2, 즉, (Gly2Ser)n (여기서, n은 1 이상의 양의 정수임)을 포함한다. 예를 들어, n=1, n=2, n=3, n=4, n=5 및 n=6, n=7, n=8, n=9 및 n=10이다. 한 실시양태에서, 링커는 (Gly4Ser)4 또는 (Gly4Ser)3을 포함하고 이로 제한되지 않는다. 또 다른 실시양태에서, 링커 Glu 및 Lys 잔기가 보다 우수한 용해도를 위해 Gly-Ser 링커 내에 산재된다. 또 다른 실시양태에서, 링커는 (Gly2Ser), (GlySer) 또는 (Gly3Ser)의 다수의 반복체를 포함한다. 또 다른 실시양태에서, 링커는 (Gly3Ser)+(Gly4Ser)+(GlySer)의 조합 및 병렬을 포함한다. 또 다른 실시양태에서, Ser은 Ala으로 교체, 예를 들어, (Gly4Ala) 또는 (Gly3Ala)일 수 있다. 또 다른 실시양태에서, 링커는 Gly, Ser 및 Pro의 임의의 조합을 포함한다. 또 다른 실시양태에서, 링커는 모티프 (GluAlaAlaAlaLys)n를 포함하고, n은 1 이상의 양의 정수이다.
본원에서 사용되는 용어 "Fc 영역"은 CH3, CH2 및 항체의 불변 도메인의 힌지 영역의 적어도 일부를 포함하는 폴리펩티드를 의미한다. 임의로, Fc 영역은 몇몇의 항체 클래스에 존재하는 CH4 도메인을 포함할 수 있다. Fc 영역은 항체의 불변 도메인의 전체 힌지 영역을 포함할 수 있다. 한 실시양태에서, 본 발명은 항체의 Fc 영역 및 CH1 영역을 포함한다. 한 실시양태에서, 본 발명은 항체의 Fc 영역 및 CH3 영역을 포함한다. 또 다른 실시양태에서, 본 발명은 Fc 영역, CH1 영역 및 항체의 불변 도메인으로부터의 C카파/람다 영역을 포함한다. 한 실시양태에서, 본 발명의 결합 분자는 불변 영역, 예를 들어, 중쇄 불변 영역을 포함한다. 한 실시양태에서, 상기 불변 영역은 야생형 불변 영역에 비해 변형된다. 즉, 본원에 개시된 본 발명의 폴리펩티드는 3개의 중쇄 불변 도메인 (CH1, CH2 또는 CH3) 중의 하나 이상 및 또는 경쇄 불변 영역 도메인 (CL)에 대한 변경 또는 변형을 포함할 수 있다. 예시적인 변형은 하나 이상의 도메인 내의 하나 이상의 아미노산의 부가, 결실 또는 치환을 포함한다. 상기 변화는 이펙터 기능, 반감기 등을 최적화하기 위해 포함될 수 있다.
본원에서 사용되는 용어 "결합 부위"는 항체 또는 항원 결합 단편이 선택적으로 결합하는 표적 수용체 상의 영역을 포함한다. 예를 들어, LRP6 상의 결합 부위는 β-프로펠러 1 결합 도메인, β-프로펠러 2 결합 도메인, β-프로펠러 3 결합 도메인, 및 β-프로펠러 4 결합 도메인을 포함한다.
본원에서 사용되는 용어 "에피토프"는 이뮤노글로불린에 고친화도로 결합할 수 있는 임의의 결정인자를 의미한다. 에피토프는 항원을 특이적으로 표적으로 하는 항체에 의해 결합되는 항원의 영역이고, 항원이 단백질인 경우, 항체와 직접 접촉하는 특이적 아미노산을 포함한다. 가장 흔하게는, 에피토프는 단백질 상에 존재하지만, 몇몇 경우에는 다른 종류의 분자, 예컨대 핵산 상에 존재할 수 있다. 에피토프 결정인자는 아미노산, 당 측쇄, 포스포릴 또는 술포닐 기와 같은 분자들의 화학적으로 활성인 표면 기를 포함할 수 있고, 특이적인 3차원 구조 특성 및/또는 특이적인 전하 특성을 가질 수 있다.
일반적으로, 특정 표적 항원에 특이적인 항체는 단백질 및/또는 거대분자의 복합 혼합물 내의 표적 항원 상의 에피토프에 결합할 것이다.
에피토프를 포함하는 제시된 폴리펩티드의 영역은 당업계에 잘 공지된 임의의 많은 에피토프 맵핑 (mapping) 기술을 사용하여 확인할 수 있다. 예를 들어, 문헌 [Epitope Mapping Protocols in Methods in Molecular Biology, Vol. 66 (Glenn E. Morris, Ed., 1996) Humana Press, Totowa, New Jersey] 참조. 예를 들어, 선형 에피토프는 예를 들어 단백질 분자의 일부분들에 대응하는 매우 많은 펩티드를 고체 지지체 상에 동시에 합성하고 펩티드가 지지체 상에 계속 부착된 상태에서 펩티드를 항체와 반응시킴으로써 결정할 수 있다. 상기 기술은 당업계에 공지되어 있고, 예를 들어, 미국 특허 번호 4,708,871; 문헌 [Geysen et al., (1984) Proc. Natl. Acad. Sci. USA 8:3998-4002]; [Geysen et al., (1985) Proc. Natl. Acad. Sci. USA 82:78-182]; [Geysen et al., (1986) Mol. Immunol. 23:709-715]에 기재되어 있다. 이와 유사하게, 입체형태적 에피토프는 예를 들어 x-선 결정학 및 2차원 핵 자기 공명에 의해 아미노산의 공간 입체형태를 결정함으로써 쉽게 확인된다. 예를 들어, 문헌 [Epitope Mapping Protocols, 상기 문헌] 참조. 또한, 단백질의 항원 영역은 표준 항원성 및 친수도 도표 (hydropathy plot)를 사용하여 확인될 수 있고, 예컨대 옥스포드 몰레큘라 그룹 (Oxford Molecular Group)으로부터 입수가능한 오미가 (Omiga) 버전 1.0 소프트웨어 프로그램을 사용하여 계산되는 것이다. 상기 컴퓨터 프로그램은 항원성 프로파일 결정을 위해 호프/우즈 (Hopp/Woods) 방법 (Hopp et al., (1981) Proc. Natl. Acad. Sci USA 78:3824-3828); 및 친수도 도표를 위해 카이트-둘리틀 (Kyte-Doolittle) 기술 (Kyte et al., (1982) J.MoI. Biol. 157:105-132)을 이용한다.
용어 2개의 엔티티 (entity) 사이의 "특이적 결합"은 적어도 102M-1, 적어도 5x102M-1, 적어도 103M-1, 적어도 5x103M-1, 적어도 104M-1, 적어도 5x104M-1, 적어도 105M-1, 적어도 5x105M-1, 적어도 106M-1, 적어도 5x106M-1, 적어도 107M-1, 적어도 5x107M-1, 적어도 108M-1, 적어도 5x108M-1, 적어도 109M-1, 적어도 5x109M-1, 적어도 1010M-1, 적어도 5x1010M-1, 적어도 1011M-1, 적어도 5x1011M-1, 적어도 1012M-1, 적어도 5x1012M-1, 적어도 1013M-1, 적어도 5x1013M-1, 적어도 1014M-1, 적어도 5x1014M-1, 적어도 1015M-1, 또는 적어도 5x1015M-1의 평형 상수 (KA) (kon/koff)로의 결합을 의미한다.
항체 (예를 들어, LRP6 항체)에 "특이적으로 (또는 선택적으로) 결합하다"는 구문은 단백질 및 다른 생물학적 물질의 불균일 집단 내의 동족 항원 (예를 들어, 인간 LRP6)의 존재를 결정하는 결합 반응을 의미한다. 상기한 평형 상수 (KA) 이외에, 본 발명의 LRP6 항체는 또한 일반적으로 5x10-2M 미만, 10-2M 미만, 5x10-3M 미만, 10-3M 미만, 5x10-4M 미만, 10-4M 미만, 5x10-5M 미만, 10-5M 미만, 5x10-6M 미만, 10-6M 미만, 5x10-7M 미만, 10-7M 미만, 5x10-8M 미만, 10-8M 미만, 5x10-9M 미만, 10-9M 미만, 5x10-10M 미만, 10-10M 미만, 5x10-11M 미만, 10-11M 미만, 5x10-12M 미만, 10-12M 미만, 5x10-13M 미만, 10-13M 미만, 5x10-14M 미만, 10-14M 미만, 5x10-15M 미만, 10-15M 미만, 또는 그 미만의 해리 속도 상수 (KD) (koff/kon)를 갖고, 비특이적 항원 (예를 들어, HSA)에 대한 결합을 위한 그의 친화도보다 적어도 2배 더 큰 친화도로 LRP6에 결합한다. 한 실시양태에서, LRP6 항체는 본원에서 설명되거나 당업자에 공지된 방법 (예를 들어, 비아코어 검정, ELISA, FACS, SET) (비아코어 인터내셔널 에이비 (비아코어 International AB, 스웨덴 웁살라))을 사용하여 평가시에 3000 pM 미만, 2500 pM 미만, 2000 pM 미만, 1500 pM 미만, 1000 pM 미만, 750 pM 미만, 500 pM 미만, 250 pM 미만, 200 pM 미만, 150 pM 미만, 100 pM 미만, 75 pM 미만, 10 pM 미만, 1 pM 미만의 해리 상수 (Kd)를 갖는다.
본원에서 사용된 바와 같이, 용어 "K회합" 또는 "Ka"는 특정 항체-항원 상호작용의 결합 속도를 지칭하는 것이고, 본원에서 사용된 바와 같이 용어 "K해리" 또는 "Kd"는 특정 항체-항원 상호작용의 해리 속도를 의미한다. 본원에서 사용된 바와 같이, 용어 "KD"는 해리 상수를 지칭하기 위한 것이고, Ka에 대한 Kd의 비율 (즉, Kd/Ka)로부터 구하며, 몰 농도 (M)로 표시된다. 항체에 대한 KD 값은 당업계에 널리 확립된 방법을 이용하여 결정할 수 있다. 항체의 KD를 결정하는 방법은 표면 플라즈몬 공명을 이용하거나 바이오센서 시스템, 예컨대 비아코어® 시스템을 이용하는 것이다.
본원에서 사용되는 용어 "친화도"는 단일 항원성 부위에서 항체와 항원 사이의 상호작용의 강도를 의미한다. 각각의 항원 부위 내에서, 항체 "아암"의 가변 영역은 많은 부위에서 약한 비-공유 힘을 통해 항원과 상호작용하고, 상호작용이 많을수록 친화도가 더 강하다.
본원에서 사용되는 용어 "결합력"은 항체-항원 복합체의 전체 안정성 또는 강도에 관한 유용한 척도를 의미한다. 이는 항체 에피토프 친화도, 항원과 항체 둘 다의 결합가, 및 상호작용 부분의 구조적 배렬이라는 3가지 주요 인자에 의해 제어된다. 궁극적으로, 이들 인자는 항체의 특이성, 즉, 특정 항체가 정확한 항원 에피토프에 결합하는 가능성을 규정한다.
본원에서 사용되는 구문 "길항제 항체"는 LRP6에 결합하고 정형적 Wnt 신호 전달의 생물학적 활성을 저하시키는, 예를 들어 Wnt 리포터 유전자 검정, 또는 포스포 LRP6 검정에서 LRP6 유도 신호 전달 활성을 저하, 감소 및/또는 억제하는 항체를 의미한다. 검정의 예를 아래 실시예에서 보다 상세히 설명한다. 몇몇 실시양태에서, 항체는 10 nM 이하, 1 nM 이하, 100 pM 이하, 10 pM, 1 pM, 0.5 pM, 0.1 pM의 IC50에서 Wnt 리포터 유전자 검정에서 측정된 LRP6 유도 활성을 저하, 감소 또는 억제한다. 몇몇 실시양태에서, 항체의 활성은 10 nM 이하, 1 nM 이하, 0.5 pM 또는 100 pM 이하의 IC50에서 SET, ELISA, FACS, 스캐챠드 (Scatchard)를 사용하여 LRP6에 대한 결합에 의해 측정될 수 있다. 한 실시양태에서, IC50은 300 μM (0.3 pM) 미만이다. 또 다른 실시양태에서, IC50은 300 μM (0.3 pM)이다. 또 다른 실시양태에서, IgG는 LRP6 Wnt의 활성을 강화한다.
본원에서 사용되는 용어 "Wnt1"은 Wnt1, Wnt2, Wnt6, Wnt7a, Wnt7b, Wnt9a, Wnt10a, 또는 Wnt10b를 의미한다.
본원에서 사용되는 용어 "Wnt3a"는 Wnt3a 및 Wnt3을 의미한다.
본원에서 사용되는 용어 "강화하다"는 Wnt 리간드의 존재 하에 항체 단편의 전장 IgG LRP6 항체로의 전환시에 Wnt 신호가 활성화되고 향상되는 과정을 의미한다.
용어 "유의한 강화 부재" 또는 "강화를 방지하다"는 Wnt 신호가 동일한 에피토프에 결합하는 대조군 항체 또는 그의 단편에 비해 활성화되거나 향상되지 않음을 나타낸다. 유의한 강화 부재는 대조군 항체 또는 그의 단편보다 적어도 10% 더 작거나, 대조군 항체 또는 그의 단편보다 적어도 20%, 적어도 30%, 적어도 40%, 적어도 50%, 적어도 60%, 적어도 70%, 적어도 80%, 적어도 90%, 적어도 100% 더 작음을 나타낼 수 있다.
본원에서 사용되는 용어 "클러스터"는 LRP6 수용체를 함께 모으거나 무리를 짓게 하고 Wnt 신호 전달을 강화하는 임의의 단백질을 의미한다. 상기 단백질의 예는 Wnt1 리간드, Wnt3a 리간드 및 Wnt3 리간드를 포함하고 이로 제한되지 않는다. 상기 단백질은 2개의 내인성 LRP6 수용체의 다량체화, 예를 들어 이량체화를 야기할 수 있다. 상기 이량체화는 LRP6의 증가된 상호작용에 의해 결합력을 증가시킬 수 있고, Wnt 리간드의 존재 하에 Wnt 신호를 강화할 수 있다.
본원에서 사용되는 구문 "단리된 항체"는 상이한 항원 특이성을 갖는 다른 항체가 실질적으로 없는 항체를 의미한다 (예를 들어, LRP6에 특이적으로 결합하는 단리된 항체에는 LRP6 이외의 항원에 특이적으로 결합하는 항체가 실질적으로 없음). 그러나, LRP6에 특이적으로 결합하는 단리된 항체는 다른 항원에 대한 교차 반응성을 가질 수 있다. 추가로, 단리된 항체에는 다른 세포 물질 및/또는 화학물질이 실질적으로 없을 수 있다.
구문 "보존적으로 변형된 변이체"는 아미노산 및 핵산 서열 둘 모두에 적용된다. 특정 핵산 서열의 경우, 보존적으로 변형된 변이체는 동일한 또는 본질적으로 동일한 아미노산 서열을 코딩하는 핵산을 지칭하거나, 또는 핵산이 아미노산 서열을 코딩하지 않는 경우에는 본질적으로 동일한 서열을 의미한다. 유전자 코드의 축퇴성 때문에, 다수의 기능적으로 동일한 핵산이 임의의 주어진 단백질을 코딩한다. 예를 들어, 코돈 GCA, GCC, GCG 및 GCU는 모두 아미노산 알라닌을 코딩한다. 따라서, 코돈에 의해 알라닌이 특정되는 모든 위치에서, 상기 코돈은 코딩되는 폴리펩티드를 변경시키지 않으면서 상기 기재된 임의의 상응하는 코돈으로 변경될 수 있다. 이러한 핵산 변이는 보존적으로 변형된 변이 중 하나인 "침묵 변이"이다. 또한, 폴리펩티드를 코딩하는 본원에서의 모든 핵산 서열은 핵산의 모든 가능한 침묵 변이도 설명한다. 당업자는 핵산 내의 각각의 코돈 (통상적으로 메티오닌에 대한 유일한 코돈인 AUG, 및 통상적으로 트립토판에 대한 유일한 코돈인 TGG 제외)이 변형되어 기능적으로 동일한 분자를 생성할 수 있음을 인식할 것이다. 따라서, 폴리펩티드를 코딩하는 핵산의 각각의 침묵 변이는 각각의 기재된 서열에 내포된다.
폴리펩티드 서열의 경우, "보존적으로 변형된 변이체"는 아미노산을 화학적으로 유사한 아미노산으로 치환시키는, 폴리펩티드 서열에 대한 개별적인 치환, 결실 또는 부가를 포함한다. 기능적으로 유사한 아미노산을 제공하는 보존적 치환 표는 당업계에 공지되어 있다. 이러한 보존적으로 변형된 변이체는 본 발명의 다형성 변이체, 종간 상동체 및 대립유전자에 부가적인 것이고 이것들을 제외하지 않는다. 다음과 같은 8개의 군은 서로에 대해 보존적 치환인 아미노산을 함유한다: 1) 알라닌 (A), 글리신 (G), 2) 아스파르트산 (D), 글루탐산 (E), 3) 아스파라긴 (N), 글루타민 (Q), 4) 아르기닌 (R), 리신 (K), 5) 이소류신 (I), 류신 (L), 메티오닌 (M), 발린 (V), 6) 페닐알라닌 (F), 티로신 (Y), 트립토판 (W), 7) 세린 (S), 트레오닌 (T), 및 8) 시스테인 (C), 메티오닌 (M) (예를 들어, 문헌 [Creighton, Proteins (1984)] 참조). 일부 실시양태에서, 용어 "보존적 서열 변형"은 아미노산 서열을 함유하는 항체의 결합 특징에 유의하게 영향을 미치거나 이를 변경하지 않는 아미노산 변형을 지칭하기 위해 사용된다.
용어 "교차-차단", "교차-차단된" 및 "교차-차단성"은 본원에서 표준 경쟁적 결합 검정에서 다른 항체 또는 결합제의 LRP6에 대한 결합을 저해하는 항체 또는 다른 결합제의 능력을 의미하기 위해 본원에서 교환가능하게 사용된다.
LRP6에 대한 또 다른 항체 또는 결합 분자의 결합을 저해할 수 있는 항체 또는 다른 결합제의 능력 또는 정도, 및 그에 따라 이것이 본 발명에 따른 교차-차단이라 할 수 있는지의 여부는 표준 경쟁 결합 검정을 이용하여 결정할 수 있다. 적합한 검정 중 하나는 표면 플라즈몬 공명 기술을 이용하여 상호작용의 정도를 측정할 수 있는 비아코어 기술 (예를 들어 비아코어 3000 기기 (비아코어, 스웨덴 웁살라)를 사용함)의 이용을 포함한다. 교차-차단을 측정하기 위한 또 다른 검정은 ELISA-기반 방법을 이용한다.
본원에서 사용되는 용어 "최적화된""은, 뉴클레오티드 서열이 생성 세포 또는 유기체, 일반적으로는 진핵 세포, 예를 들어 피치아 (Pichia)의 세포, 트리코더마 (Trichoderma)의 세포, 차이니즈 햄스터 난소 세포 (CHO) 또는 인간 세포에 바람직한 코돈을 사용하여 아미노산 서열을 코딩하도록 변경된 것을 의미한다. 최적화된 뉴클레오티드 서열은 "모" 서열이라고도 알려진 출발 뉴클레오티드 서열에 의해 원래 코딩되는 아미노산 서열을 완전히 또는 가능한 많이 보유하도록 조작된다.
다양한 종의 LRP6에 대한 항체의 결합 능력을 평가하기 위한 표준 검정, 예를 들어, ELISA, 웨스턴 블롯 및 RIA가 당업계에 공지되어 있다. 적합한 검정은 실시예에서 상세히 설명한다. 또한, 항체의 결합 동역학 (예를 들어, 결합 친화도)은 당업계에 공지된 표준 검정, 예컨대 비아코어 분석, 또는 FACS 상대 친화도 (스캐챠드)에 의해 평가될 수 있다. LRP6의 기능적 특성에 대한 항체의 효과를 평가하기 위한 검정 (예를 들어, Wnt 경로를 조정하는 수용체 결합 검정)은 실시예에서 추가로 상세히 설명한다.
따라서, 당업계에 공지되고 본원에서 설명되는 방법에 따라 결정되는 바와 같이 하나 이상의 상기 LRP6 기능적 특성 (예를 들어, 생화학적, 면역화학적, 세포성, 생리학적 또는 다른 생물학적 활성 등)을 "억제하는" 항체는 항체의 부재 하에 (예를 들어, 또는 무관한 특이성의 대조군 항체가 존재할 때) 관찰되는 것에 비해 특정 활성의 통계적으로 유의한 감소와 관련되는 것으로 이해될 것이다. LRP6 활성을 억제하는 항체는 측정된 파라미터를 적어도 10%, 적어도 50%, 80% 또는 90%까지 상기 통계적으로 유의한 감소를 초래하고, 특정 실시양태에서 본 발명의 항체는 LRP6 기능적 활성을 95%, 98% 또는 99%보다 큰 수준으로 억제할 수 있다.
2개 이상의 핵산 또는 폴리펩티드 서열과 관련하여 용어 "동일한 %" 또는 "동일성 %"는 동일한 2개 이상의 서열 또는 하위서열을 의미한다. 2개의 서열은, 비교 범위 또는 지정된 영역에 걸쳐 최대한 대응하도록 비교하고 정렬하여 하기하는 서열 비교 알고리즘 중 하나를 이용하거나 또는 수동 정렬 및 시각적 검사에 의해 결정하는 경우, 2개의 서열이 명시된 백분율의 동일한 아미노산 잔기 또는 뉴클레오티드 (즉, 명시된 영역에 걸쳐서 또는 명시되지 않은 경우에는 전체 서열에 걸쳐서 60%의 동일성, 임의로 65%, 70%, 75%, 80%, 85%, 90%, 95% 또는 99%의 동일성)를 가질 경우에 "실질적으로 동일한" 것이다. 임의로, 동일성은 적어도 약 50개 뉴클레오티드 (또는 10개 아미노산) 길이의 영역에 걸쳐, 보다 바람직하게는 100개 내지 500개 또는 1000개 또는 그보다 많은 수의 뉴클레오티드 (또는 20개, 50개, 200개 또는 그보다 많은 수의 아미노산) 길이에 걸쳐 존재한다.
서열 비교를 위해, 일반적으로 하나의 서열이 시험 서열과 비교되는 참조 서열로서 작용한다. 서열 비교 알고리즘을 이용하는 경우, 시험 및 참조 서열을 컴퓨터에 입력하고, 필요한 경우에는 하위서열 좌표를 지정하고 서열 알고리즘 프로그램 파라미터를 지정한다. 디폴트 프로그램 파라미터가 사용될 수도 있거나, 또는 대안적인 파라미터가 지정될 수도 있다. 이후, 서열 비교 알고리즘은 프로그램 파라미터를 기초로 하여 참조 서열에 대한 시험 서열의 서열 동일성 %를 계산한다.
본원에서 사용된 바와 같이, "비교 범위"는 2개의 서열을 최적으로 정렬시킨 후에 인접 위치의 동일한 수의 참조 서열과 그 서열을 비교할 수 있는, 20개 내지 600개, 일반적으로는 약 50개 내지 약 200개, 보다 일반적으로는 약 100개 내지 약 150개로 이루어진 군 중에서 선택된 인접 위치의 수 중 임의의 하나의 절편에 대한 것을 포함한다. 비교를 위한 서열 정렬의 방법은 당업계에 공지되어 있다. 비교를 위한 서열의 최적 정렬은 예를 들어 문헌 [Smith and Waterman (1970) Adv. Appl. Math. 2:482c]의 국소 상동성 알고리즘, 문헌 [Needleman and Wunsch (1970) J. Mol. Biol. 48:443]의 상동성 정렬 알고리즘, 문헌 [Pearson and Lipman, (1988) Proc. Nat'l. Acad. Sci. USA 85:2444]의 유사성 검색 방법, 이들 알고리즘의 전산화 실행 (위스콘신 제네틱스 소프트웨어 패키지 (Wisconsin Genetics Software Package)의 GAP, BESTFIT, FASTA 및 TFASTA, 제네틱스 컴퓨터 그룹 (Genetics Computer Group) (미국 위스콘신주 메디슨 사이언스 드라이브 575)) 또는 수동 정렬 및 시각적 검사 (예를 들어, 문헌 [Brent et al., (2003) Current Protocols in Molecular Biology] 참조)에 의해 수행될 수 있다.
서열 동일성 및 서열 유사성 % 결정에 적합한 알고리즘의 2가지 예는 각각 문헌 [Altschul et al., (1977) Nuc. Acids Res. 25:3389-3402] 및 [Altschul et al., (1990) J. Mol. Biol. 215:403-410]에 기재된 BLAST 및 BLAST 2.0 알고리즘이다. BLAST 분석을 수행하기 위한 소프트웨어는 미국 국립 생물공학 정보 센터 (National Center for Biotechnology Information)를 통해 공개적으로 입수가능하다. 이러한 알고리즘은 먼저 데이터베이스 서열 내 동일 길이의 워드 (word)와 정렬될 때 약간의 양성 값의 역치 스코어 T에 매칭되거나 이를 충족시키는, 질의 (query) 서열 내의 길이 W의 짧은 워드들을 확인함으로써 높은 스코어의 서열 쌍 (HSP)을 확인하는 것을 포함한다. T는 이웃 워드 스코어 역치를 의미한다 ([Altschul et al., 상기 문헌]). 이러한 초기 이웃 워드 적중치 (hit)는 이것을 함유하는 더 긴 HSP를 찾기 위한 검색을 시작하기 위한 시드 (seed)로 작용한다. 워드 적중치는 누적 정렬 스코어가 증가될 수 있는 한 각각의 서열을 따라 양방향으로 멀리 연장된다. 누적 스코어는 뉴클레오티드 서열에 대해 파라미터 M (매치 잔기의 쌍에 대한 보상 스코어, 항상 > 0) 및 N (미스매치 잔기에 대한 패널티 스코어, 항상 < 0)을 사용하여 계산한다. 아미노산 서열의 경우, 누적 스코어 계산에 스코어링 매트릭스가 사용된다. 누적 정렬 스코어가 이의 최대 달성 값으로부터 X의 양만큼 하락한 경우, 1개 이상의 음의 값으로 스코어링된 잔기 정렬의 축적으로 인해 누적 스코어가 0 이하가 된 경우, 또는 어느 한쪽 서열의 끝에 도달한 경우에는 각 방향으로의 워드 적중치의 연장이 중단된다. BLAST 알고리즘 파라미터 W, T 및 X는 정렬의 감도 및 속도를 결정한다. BLASTN 프로그램 (뉴클레오티드 서열의 경우)은 디폴트로서 워드 길이 (W) 11, 기대값 (E) 10, M = 5, N = -4 및 양가닥의 비교를 사용한다. 아미노산 서열의 경우, BLASTP 프로그램은 디폴트로서 워드 길이 3, 및 기대값 (E) 10, 및 BLOSUM62 스코어링 매트릭스 (문헌 [Henikoff and Henikoff, (1989) Proc. Natl. Acad. Sci. USA 89:10915] 참조) 정렬 (B) 50, 기대값 (E) 10, M = 5, N = -4, 및 양가닥의 비교를 사용한다.
BLAST 알고리즘은 또한 2개의 서열 사이의 유사성에 대한 통계학적 분석을 수행한다 (예를 들어, 문헌 [Karlin and Altschul (1993) Proc. Natl. Acad. Sci. USA 90:5873-5787] 참조). BLAST 알고리즘에 의해 제공되는 유사성의 한 척도는 2개의 뉴클레오티드 또는 아미노산 서열 사이의 매치가 우연히 발생할 확률을 나타내는, 최소의 합계 확률 (P(N))이다. 예를 들어, 핵산은 참조 핵산에 대한 시험 핵산의 비교시에 최소의 합계 확률이 약 0.2 미만, 보다 바람직하게는 약 0.01 미만, 가장 바람직하게는 약 0.001 미만일 경우에 참조 서열과 유사한 것으로 간주된다.
또한, 2개의 아미노산 서열 사이의 동일성 %는, PAM120 가중 잔기 표, 갭 (gap) 길이 패널티 12 및 갭 패널티 4를 사용하여 ALIGN 프로그램 (버전 2.0)에 도입된 이. 메이어스 (E. Meyers) 및 더블유. 밀러 (W. Miller)의 알고리즘 (Comput. Appl. Biosci. 4:11-17 (1988))을 이용하여 결정할 수 있다. 또한, 2개의 아미노산 서열 사이의 동일성 %는 GCG 소프트웨어 패키지 (www.gcg.com에서 이용가능함) 내의 GAP 프로그램에 도입된 니들만 (Needleman) 및 분쉬 (Wunsch) (J. Mol, Biol. 48:444-453 (1970)) 알고리즘을 이용하여, 블로섬 (Blossom) 62 매트릭스 또는 PAM250 매트릭스, 및 갭 가중치 16, 14, 12, 10, 8, 6 또는 4, 및 길이 가중치 1, 2, 3, 4, 5 또는 6을 사용하여 결정할 수 있다.
상기 나타낸 서열 동일성 백분율 이외에, 2개의 핵산 서열 또는 폴리펩티드가 실질적으로 동일하다는 또 다른 표시는 제1 핵산에 의해 코딩되는 폴리펩티드가 하기하는 바와 같이 제2 핵산에 의해 코딩되는 폴리펩티드에 대해 생성된 항체와 면역학적으로 교차 반응성이라는 것이다. 따라서, 폴리펩티드는 예를 들어 2개의 펩티드가 보존적 치환에 의해서만 상이한 경우에 일반적으로 제2 폴리펩티드에 실질적으로 동일하다. 2개의 핵산 서열이 실질적으로 동일함을 보여주는 또 다른 표시는 2개의 분자 또는 이것들의 상보체가 하기하는 바와 같이 엄격한 조건 하에 서로 혼성화된다는 것이다. 2개의 핵산 서열이 실질적으로 동일함을 보여주는 또 다른 표시는 동일한 프라이머가 서열 증폭에 사용될 수 있다는 것이다.
구문 "핵산"은 본원에서 용어 "폴리뉴클레오티드"와 교환가능하게 사용되고, 단일 가닥 또는 이중 가닥 형태의 데옥시리보뉴클레오티드 또는 리보뉴클레오티드 및 그의 중합체를 의미한다. 상기 용어는 공지의 뉴클레오티드 유사체 또는 변형된 주쇄 잔기 또는 연결부를 함유하고, 합성, 천연 생성 및 비-천연 생성이며, 참조 핵산과 유사한 결합 특성을 가지며, 참조 뉴클레오티드와 유사한 방식으로 대사되는 핵산을 포함한다. 이러한 유사체의 예는 포스포로티오에이트, 포스포르아미데이트, 메틸 포스포네이트, 키랄-메틸 포스포네이트, 2-O-메틸 리보뉴클레오티드, 펩티드-핵산 (PNA)을 포함하고 이로 제한되지 않는다.
달리 언급하지 않는 한, 특정 핵산 서열은 또한 명시적으로 나타낸 서열뿐만 아니라 그의 보존적으로 변형된 변이체 (예를 들어, 축퇴성 코돈 치환) 및 상보성 서열도 함축적으로 포함한다. 구체적으로, 아래에서 상세하게 설명된 바와 같이, 축퇴성 코돈 치환은 1개 이상의 선택된 (또는 모든) 코돈의 제3의 위치가 혼합-염기 및/또는 데옥시이노신 잔기로 치환된 서열을 생성시켜 달성될 수 있다 ([Batzer et al., (1991) Nucleic Acid Res. 19:5081]; [Ohtsuka et al., (1985) J. Biol. Chem. 260:2605-2608]; 및 [Rossolini et al., (1994) Mol. Cell. Probes 8:91-98]).
용어 "작동가능하게 연결된"은 2개 이상의 폴리뉴클레오티드 (예를 들어, DNA) 절편 사이의 기능적 관계를 의미한다. 일반적으로, 이것은 전사되는 서열에 대한 전사 조절 서열의 기능적 관계를 의미한다. 예를 들어, 프로모터 또는 인핸서 서열이 적절한 숙주 세포 또는 다른 발현 시스템에서 코딩 서열의 전사를 자극하거나 조정하는 경우, 이것은 코딩 서열에 작동가능하게 연결된 것이다. 일반적으로, 전사되는 서열에 작동가능하게 연결된 프로모터 전사 조절 서열은 전사되는 서열에 물리적으로 인접하여 위치하는데, 즉, 이것들은 시스-작용성 (cis-acting)이다. 그러나, 일부 전사 조절 서열, 예컨대 인핸서는 이것이 전사를 증진시키는 코딩 서열에 물리적으로 인접하거나 근접하여 위치할 필요가 없다.
용어 "폴리펩티드" 및 "단백질"은 본원에서 교환가능하게 사용되고, 아미노산 잔기의 중합체를 의미한다. 상기 용어는 천연 생성 아미노산 중합체 및 비-천연 생성 아미노산 중합체뿐만 아니라 1개 이상의 아미노산 잔기가 상응하는 천연 생성 아미노산의 인공적인 화학적 모방체인 아미노산 중합체에도 적용된다. 달리 언급하지 않는 한, 특정 폴리펩티드 서열은 또한 그의 보존적으로 변형된 변이체도 함축적으로 포함한다.
용어 "대상체"는 인간 및 비-인간 동물을 포함한다. 비-인간 동물은 모든 척추동물, 예를 들어 포유동물 및 비-포유동물, 예컨대 비-인간 영장류, 양, 개, 소, 닭, 양서류 및 파충류를 포함한다. 명시되는 경우를 제외하고는, 용어 "환자" 또는 "대상체"는 본원에서 교환가능하게 사용된다.
용어 "항암제"는 세포독성제, 화학치료제, 방사선 요법 및 방사선치료제, 표적 지정 항암제, 및 면역치료제를 비롯하여, 세포 증식성 장애, 예컨대 암을 치료하기 위해 사용될 수 있는 임의의 작용제를 의미한다.
"종양"은 신생물성 세포 성장 및 증식 (악성 또는 양성 불문), 및 모든 전암성 및 암성 세포 및 조직을 의미한다.
용어 "항-종양 활성"은 종양 세포 증식, 생존력, 또는 전이 활성의 비율의 감소를 의미한다. 항-종양 활성을 보여주는 가능한 방식은 요법 동안 비정상적인 세포의 성장 속도의 감소 또는 종양 크기 안정성 또는 감소를 보여주는 것이다. 상기 활성은 이종이식편 모델, 동종이식편 모델, MMTV 모델, 및 항-종양 활성을 조사하기 위한 당업계에 공지된 다른 모델을 포함하고 이로 제한되지 않는, 승인된 시험관내 또는 생체내 종양 모델을 사용하여 평가될 수 있다.
용어 "악성종양"은 비-양성 종양 또는 암을 의미한다. 본원에서 사용되는 바와 같이, 용어 "암"은 탈조절되거나 비제어된 세포 성장을 특징으로 하는 악성종양을 포함한다. 예시적인 암은 암종, 육종, 백혈병, 및 림프종을 포함한다. 용어 "암"은 원발성 악성 종양 (예를 들어, 그 세포가 대상체의 본래 종양의 부위 이외의 다른 신체 내의 부위로 이동하지 않은 종양) 및 2차 악성 종양 (예를 들어, 전이, 즉 종양 세포가 본래 종양의 부위와 상이한 2차 부위로 이동함으로써 발생하는 종양)을 포함한다.
본 발명의 다양한 측면은 다음 섹션 및 하위섹션에서 추가로 상세히 설명한다.
LRP6
및
Wnt
-신호 전달 경로
본 발명은 LRP6 항체 및 그의 용도에 관한 것이다. LRP6에 대해 작용하는 분자에 의한 Wnt 신호 전달의 억제는 정형적 Wnt 신호 전달의 상실을 유발한다. 따라서, 항체를 사용한 LRP6 수용체 기능의 길항작용은 Wnt 신호 전달을 억제하고, 정형적 Wnt 신호 전달과 연관된 질환, 예를 들어, 암에 대해 도움을 줄 것이다. 특히, LRP6 항체는 상이한 질환 상황에서 Wnt1 또는 Wnt3a 클래스 단백질에 의해 매개되는 신호 전달을 특이적으로 증가 또는 감소시킬 수 있다.
Wnt/β-카테닌 신호 전달 경로의 오조절은 다양한 인간 질환, 예컨대 암 및 골 장애와 연결된다. 상기 질환에서 Wnt 신호 전달의 균형을 회복시키는 분자는 치료 능력을 가질 수 있다. 파지-기반 패닝 (panning)을 이용하여, Wnt 신호 전달을 억제 또는 향상시키는 LRP6 항체를 확인하였다. 특이하게도, LRP6 길항 항체의 2개의 클래스가 확인되었다. 항체의 한 클래스는 Wnt1로 제시되는 Wnt 단백질을 특이적으로 억제하는 반면에, 제2 클래스는 Wnt3a으로 제시되는 Wnt 단백질을 특이적으로 억제한다. 에피토프 맵핑 실험은 Wnt1-특이적 및 Wnt3a-특이적 LRP6 항체가 각각 LRP6의 제1 프로펠러 및 제3 프로펠러에 결합함을 나타내고, 이것은 Wnt1 및 Wnt3a 단백질이 LRP6의 상이한 프로펠러 영역에 결합함을 시사한다 (그 내용 전체가 본원에 참고로 포함되는 국제 출원 일련 번호 PCT/EP2008/064821 (2008년 10월 31일 출원) 참조).
LRP6의 프로펠러 3 도메인에 대한 추가의 특징 결정을 통해 항체와의 상호작용을 담당하는 상기 도메인 내의 잔기를 확인하였다. 프로펠러 3의 YWTD-EGF 영역 내의 항체 결합 부위는 수소-중수소 교환 (HDx) 질량 분광법 (MS)을 사용하여 확인하였고, 프로펠러 3 도메인의 날개깃 1과 6 사이의 오목한 표면에 대응한다.
Wnt 신호 전달 경로는 배 발생 및 출산후 조직 유지에 중요하다. 이것은 세포 성장의 일시적인 및 공간적 조절, 이동 및 세포 생존을 제어하는 유전자의 특이적 세트를 유도함으로써 달성된다 (문헌 [Barker and Clevers (2006) Nature Rev. 5:997]에서 검토됨). 상기 경로의 적절한 조절은 조직 항상성 유지에 중요하다. 상기 경로의 만성 활성화는 비제어된 세포 성장 및 생존을 촉진하고, 결과적으로 세포 증식성 질환, 예컨대 암의 발생을 유도할 수 있다. 별법으로, 상기 경로의 비정상적인 억제는 많은 질환 상태, 예를 들어 골 질량의 손실 및 다른 골 질환을 야기할 수 있다. Wnt 단백질은 프리즐드 수용체, 및 저밀도 지단백질 수용체 (LDLR)-관련 단백질 (LRP)의 멤버인 2개의 세포-표면 수용체 LRP5 및 LRP6 중의 하나와 상호작용함으로써 하류의 신호 전달을 개시시킨다 (문헌 [He et al., (2004) Development 31:1663-1677]에서 검토됨).
정형적 Wnt 신호 전달에서 LRP6의 역할은 유전적 연구를 통해 밝혀졌다. LRP6이 결여된 돌연변이체 마우스는 몇몇의 개별적인 Wnt 유전자 내의 돌연변이와 유사한 복합적인 표현형을 보였다 (Pinson et al., (2000) Nature 407:535-538). 제노푸스 (Xenopus) 배아에서, 우성-음성 LRP6은 몇몇의 Wnt 단백질에 의한 신호 전달을 차단한 반면, LRP6의 과다발현은 Wnt/β-카테닌 신호 전달을 활성화시켰다 (Tamai et al., (2000) Nature 407:530-535). 또한, 세포가 정형적 Wnt 신호 전달에 반응하기 위해 LRP6 또는 LRP5의 발현이 필요함이 밝혀졌다 ([He et al., 상기 문헌, 2004]에서 검토됨).
LRP5 및 LRP6은 고도로 상동성이고, 이들의 세포외 및 세포내 도메인에서 각각 73% 및 64% 동일성을 공유한다. 이들은 배아발생 동안 성체 조직에서 광범하게 동시 발현되고, 몇몇 기능이 중복된다.
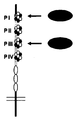
LRP5 및 LRP6의 세포외 도메인은 다음과 같은 3개의 기초적인 도메인을 포함한다: 1) YWTD (티로신, 트립토판, 트레오닌, 아스파르트산)-형 β-프로펠러 영역, 2) EGF (표피 성장 인자)-유사 도메인, 및 3) LDLR 타입 A (LA) 도메인을 포함한다.
YWTD-형 β-프로펠러 영역은 각각 43-50개 아미노산 잔기의 6개의 YWTD 반복체를 함유하고, 6-날개깃 β-프로펠러 구조를 형성한다. LRP5 및 LRP6에서, 4개의 YWTD-형 β-프로펠러 영역이 존재하고, 이어서 이들 각각의 영역 다음에 보존된 시스테인 잔기와 함께 약 40개의 아미노산 잔기를 포함하는 EGF-유사 도메인이 존재하고, 다시 3개의 LA 도메인이 존재한다 ([Springer et al., (1998) J. Mol. Biol. 283:837- 862]; [Jeon et al., (2001) Nat. Struct. Biol. 8:499-504]). β-프로펠러-EGF-유사 도메인은 세포외 리간드에 결합할 수 있다. LRP6의 세포외 도메인은 아미노산 잔기 19 내지 1246에 의해 규정되고, 각각 β-프로펠러 영역 1, 2, 3 및 4에 대응하는 아미노산 잔기 43-324, 352-627, 654-929, 및 957-1250에 4개의 β-프로펠러 도메인을 함유한다. 프로펠러 도메인 1-2는 아미노산 19-629를 포함하고, 프로펠러 도메인 3-4는 아미노산 631-1246을 포함한다.
LRP6
항체
본 발명은 LRP6 (예를 들어, 인간 LRP6, 시노몰구스 LRP6, 마우스 LRP6, 및 래트 LRP6)에 특이적으로 결합하는 항체를 제공한다. 본 발명은 β-카테닌 신호 전달을 활성화시킬 수 있는 Wnt 단백질은 2개의 클래스로 분류될 수 있고, 그 내용 전체가 본원에 참고로 포함되는 국제 출원 일련 번호 PCT/EP2008/064821 (2008년 10월 31일 출원)에 기재된 바와 같이 신호 전달을 위해 LRP6의 상이한 프로펠러를 필요로 한다는 놀라운 발견을 기초로 한다. 또한, 이량체/이가 LRP6 항체 (예를 들어, IgG)는 예를 들어 내인성 LRP6의 이량체화를 통해 세포를 Wnt 신호 전달에 대해 강하게 감수성으로 만든다. 상기 결과는 프로펠러 1 및 프로펠러 3이 Wnt1 및 Wnt3의 신호 전달 활성을 위해 차별적으로 필요함을 시사한다. 상기 발견은 Wnt-유도 LRP6 활성화에 대한 새로운 통찰력을 제공하고, 상이한 질환에서 Wnt 신호 전달을 조정하기 위한 LRP6 항체의 개발을 위한 길을 마련하였다. LRP6 항체 단편 (예를 들어, Fab)의 IgG 포맷으로의 전환은, LRP6 수용체를 모으고 리간드 단백질의 존재 하에 Wnt 신호를 강화할 수 있는 항체를 생성한다.
한 실시양태에서, 항체는 Wnt 신호를 강화한다. 상기 실시양태에서, Wnt 신호는 Wnt 리간드의 존재 하에 항체 단편의 전장 IgG LRP6 항체로의 전환시에 활성화되고 향상된다. 예를 들어, Wnt1 Fab는 LRP6 수용체의 프로펠러 1 영역에 결합하고, Wnt 리간드, 예를 들어 Wnt3의 부재 하에 Wnt1 경로를 차단한다. Wnt 리간드, 예를 들어 Wnt3의 존재 하에, Wnt1 Fab는 Wnt1 경로를 통한 신호 전달을 차단하지만, 신호 활성화는 Wnt3 경로를 통해 발생할 수 있고, 이에 의해 신호가 생성된다. Wnt1 Fab가 전장 Wnt1 IgG로 전환될 때, Wnt1 IgG는 2개의 LRP6 수용체의 프로펠러 1 영역에 결합하고, Wnt1 경로를 차단하지만, Wnt 리간드, 예를 들어 Wnt3의 존재하에; 신호 활성화는 Wnt3 경로를 통해 발생하고, 역시 향상된다. 작용 이론을 제공할 필요는 없지만, 하나의 가능한 메카니즘은 IgG가 각각의 LRP6 수용체의 프로펠러 1 영역에 결합함으로써 2개 이상의 LRP6 수용체를 함께 모으고, 이것은 Wnt3 리간드의 존재 하에 Wnt3 경로를 통한 보다 강한 신호를 생성시킨다는 것이다. LRP6 수용체의 이량체화는 아마도 LRP6을 수반하는 다양한 상호작용의 결합력 증가를 통해 Wnt 신호 전달을 촉진한다.
LRP6 수용체의 프로펠러 3 영역에 결합하고 Wnt3 경로를 차단하는 Wnt3 Fab를 사용할 경우 반대의 결과가 얻어진다. Wnt1 리간드의 존재 하에, Wnt3 Fab는 Wnt3 경로를 차단하지만, Wnt1 경로를 활성화시켜 신호를 생성한다. Wnt3 Fab가 전장 Wnt3 IgG으로 전환될 때, Wnt3 IgG는 2개의 LRP6 수용체의 프로펠러 3 영역에 결합하고, Wnt1 리간드의 존재 하에 Wnt1 경로를 통한 신호 전달을 억제한다. 또 다른 실시양태에서, 항체는 Wnt 신호의 강화를 방지한다. 몇몇 실시양태에서, 본 발명은 인간 및 시노몰구스 LRP6 둘 모두에 특이적으로 결합하는 항체를 제공한다. 한 실시양태에서, LRP6 항체는 길항 항체이다. 또 다른 실시양태에서, LRP6 항체는 효능작용성 항체이다.
상이한 Wnt 단백질은 신호 전달을 위해 LRP6의 상이한 프로펠러를 필요로 하고 LRP6의 회합 또는 이량체화는 Wnt 신호 전달을 강화하기 때문에, LRP6 항체를 사용한 요법은 항체 조합을 사용하여 조절될 수 있다.
한 실시양태에서, LRP6 항체는 단량체 항체 또는 그의 단편, 예컨대 단일 쇄 항체, 유니바디 등으로서 사용된다. 한 실시양태에서, LRP6의 프로펠러 1 영역에 결합하는 단량체 LRP6 항체는 LRP6의 프로펠러 3 영역에 결합하는 단량체 LRP6 항체와 조합되어 사용된다. 또 다른 실시양태에서, LRP6 항체는 다량체 항체 또는 그의 단편, 예컨대 이중특이적, 이중파라토프 LRP6 항체로서 사용된다.
Wnt 리간드 이외에 LRP6 프로펠러 1 항체는 다른 프로펠러 1 결합 리간드 (예를 들어 스클레로스틴, Dkk1)와의 상호작용을 억제할 것으로 예상된다. 이와 유사하게, 프로펠러 3 항체는 다른 프로펠러 3 결합 리간드 (예를 들어, Dkk1)와의 상호작용을 억제할 것으로 예상된다. 또한, 프로펠러 1 및 3 결합 항체는 다른 Wnt 신호 전달 조절물질, 예를 들어 R-스폰딘의 활성에 영향을 줄 것으로 예상할 수 있다.
또한, 본 발명은 하기 표 1에 제시된 VH CDR의 임의의 하나의 아미노산 서열을 갖는 VH CDR을 포함하는, LRP6 단백질 (예를 들어, 인간 및/또는 시노몰구스 LRP6)에 특이적으로 결합하는 항체를 제공한다. 특히, 본 발명은 하기 표 1에 제시된 임의의 VH CDR의 아미노산 서열을 갖는 1, 2, 3, 4, 5개 또는 그 초과의 VH CDR을 포함하는 (또는 별법으로, 이로 이루어지는), LRP6 단백질 (예를 들어, 인간 및/또는 시노몰구스 LRP6)에 특이적으로 결합하는 항체를 제공한다.
본 발명은 서열 14, 34, 36, 44, 60 및 62의 아미노산 서열을 갖는 VH 도메인을 포함하는, LRP6 단백질 (예를 들어, 인간 및/또는 시노몰구스 LRP6)에 특이적으로 결합하는 항체를 제공한다. 본 발명은 서열 13, 33, 35, 43, 59, 및 61의 아미노산 서열을 갖는 VL 도메인을 포함하는, LRP6 단백질 (예를 들어, 인간 및/또는 시노몰구스 LRP6)에 특이적으로 결합하는 항체를 제공한다.
본 발명은 서열 82, 89, 106, 108, 128, 130, 및 138의 아미노산 서열을 갖는 VH 도메인을 포함하는, LRP6 단백질 (예를 들어, 인간 및/또는 시노몰구스 LRP6)에 특이적으로 결합하는 항체를 제공한다. 본 발명은 서열 81, 90, 105, 107, 127, 및 129의 아미노산 서열을 갖는 VL 도메인을 포함하는, LRP6 단백질 (예를 들어, 인간 및/또는 시노몰구스 LRP6)에 특이적으로 결합하는 항체를 제공한다.
본 발명의 다른 항체는, 돌연변이되었지만 표 1에 기재된 서열에 제시된 CDR 영역과 적어도 60%, 70%, 80%, 90%, 95% 또는 98%의 CDR 영역 동일성을 갖는 아미노산을 포함한다. 몇몇 실시양태에서, 항체는 원래의 항체의 에피토프에 대한 그들의 특이성을 여전히 유지하면서, 표 1에 기재된 서열에 제시된 CDR 영역과 비교시에 1, 2, 3, 4 또는 5개의 아미노산이 CDR 영역에서 돌연변이된 돌연변이체 아미노산 서열을 포함한다.
본 발명의 다른 항체는, 돌연변이되었지만 표 1에 기재된 서열에 제시된 프레임워크 영역과 적어도 60%, 70%, 80%, 90%, 95% 또는 98%의 프레임워크 영역 동일성을 갖는 아미노산을 포함한다. 몇몇 실시양태에서, 항체는 원래의 항체의 에피토프에 대한 그들의 특이성을 여전히 유지하면서, 표 1에 기재된 서열에 제시된 프레임워크 영역과 비교시에 1, 2, 3, 4, 5, 6, 또는 7개의 아미노산이 프레임워크 영역에서 돌연변이된 돌연변이체 아미노산 서열을 포함한다.
또한, 본 발명은 LRP6 단백질 (예를 들어, 인간 및/또는 시노몰구스 LRP6)에 특이적으로 결합하는 항체의 VH, VL, 전장 중쇄, 및 전장 경쇄를 코딩하는 핵산 서열을 제공한다. 상기 핵산 서열은 포유동물 세포에서의 발현을 위해 최적화될 수 있다 (예를 들어, β-프로펠러 1 항체에 대한 MOR08168, MOR08545, 및 MOR06706 및 β-프로펠러 3 항체에 대한 MOR06475, MOR08193, 및 MOR08473에 대해 표 1 참조).
본 발명의 LRP6 항체는 구분되는 LRP6 β-프로펠러 영역에 결합한다. 프로펠러 1 항체는 β-프로펠러 1 도메인에 결합하고, 프로펠러 1-의존성 Wnt, 예컨대 Wnt1, Wnt2, Wnt6, Wnt7A, Wnt7B, Wnt9, Wnt10A, Wnt10B를 차단한다. 프로펠러 3 항체는 β-프로펠러 3 도메인에 결합하고, 프로펠러 3-의존성 Wnt, 예컨대 Wnt3a 및 Wnt3을 차단한다.
본 발명의 다른 항체는, 아미노산 또는 아미노산을 코딩하는 핵산이 돌연변이되었지만 표 1에 기재된 서열에 대해 적어도 60%, 70%, 80%, 90%, 95% 또는 98%의 동일성을 갖는 것을 포함한다. 몇몇 실시양태에서, 항체는 실질적으로 동일한 치료 활성을 보유하면서, 표 1에 기재된 서열에 제시된 가변 영역과 비교시에 1, 2, 3, 4 또는 5개 이하의 아미노산이 가변 영역에서 돌연변이된 돌연변이체 아미노산 서열을 포함한다.
각각의 이들 항체는 LRP6에 결합할 수 있기 때문에, VH, VL, 전장 경쇄, 및 전장 중쇄 서열 (아미노산 서열 및 아미노산을 코딩하는 뉴클레오티드 서열)은 본 발명의 다른 LRP6 항체를 생성하기 위해 "혼합 및 매치 (mixed and matched)"될 수 있다. 상기 "혼합 및 매치된" LRP6 항체는 당업계에 공지된 결합 검정 (예를 들어, ELISA, 및 실시예 섹션에 기재된 다른 검정)을 사용하여 시험될 수 있다. 상기 사슬이 혼합 및 매치될 때, 특정 VH/VL 페어링으로부터의 VH 서열은 구조상 유사한 VH 서열로 교체되어야 한다. 마찬가지로, 특정 전장 중쇄/전장 경쇄 페어링으로부터의 전장 중쇄 서열은 구조상 유사한 전장 중쇄 서열로 교체되어야 한다. 마찬가지로, 특정 VH/VL 페어링으로부터의 VL 서열은 구조상 유사한 VL 서열로 교체되어야 한다. 마찬가지로, 특정 전장 중쇄/전장 경쇄 페어링으로부터의 전장 경쇄 서열은 구조상 유사한 전장 경쇄 서열로 교체되어야 한다. 따라서, 한 측면에서, 본 발명은 서열 14, 34, 36, 44, 60, 및 62로 이루어진 군 중에서 선택된 아미노산 서열을 포함하는 중쇄 가변 영역; 및 서열 13, 33, 35, 43, 59 및 61로 이루어진 군 중에서 선택된 아미노산 서열을 포함하는 경쇄 가변 영역; 서열 82, 106, 108, 128, 130 및 138로 이루어진 군 중에서 선택된 중쇄; 및 서열 81, 및 90, 105, 107, 127, 129, 및 137로 이루어진 군 중에서 선택된 아미노산 서열을 포함하는 경쇄 가변 영역을 갖는 단리된 모노클로날 항체 또는 그의 단편을 제공하고; 여기서, 항체는 LRP6 (예를 들어, 인간 및/또는 시노몰구스 LRP6)에 특이적으로 결합한다.
또 다른 측면에서, 본 발명은 표 1에 기재된 중쇄 및 경쇄 CDR1, CDR2 및 CDR3, 또는 이들의 조합을 포함하는 LRP6의 β 프로펠러 1 도메인에 결합하는 LRP6 항체를 제공한다. 항체의 VH CDR1의 아미노산 서열은 서열 1, 21, 및 47에 제시된다. 항체의 VH CDR2의 아미노산 서열은 서열 2, 22, 및 48에 제시된다. 항체의 VH CDR3의 아미노산 서열은 서열 3, 23, 및 49에 제시된다. 항체의 VL CDR1의 아미노산 서열은 서열 4, 24, 및 50에 제시된다. 항체의 VL CDR2의 아미노산 서열은 서열 5, 25, 및 51에 제시된다. 항체의 VL CDR3의 아미노산 서열은 서열 6, 26, 및 52에 제시된다. CDR 영역은 카바트 시스템을 사용하여 기재된다 ([Kabat et al., (1991) Sequences of Proteins of Immunological Interest, Fifth Edition, U.S. Department of Health and Human Services, NIH Publication No. 91-3242]; [Chothia et al., (1987) J. Mol. Biol. 196: 901-917]; [Chothia et al., (1989) Nature 342: 877-883]; 및 [Al-Lazikani et al., (1997) J. Mol. Biol. 273, 927-948]).
또 다른 측면에서, 본 발명은 표 1에 기재된 중쇄 및 경쇄 CDR1, CDR2 및 CDR3, 또는 이들의 조합을 포함하는 LRP6의 β 프로펠러 3 도메인에 결합하는 LRP6 항체를 제공한다. 항체의 VH CDR1의 아미노산 서열은 서열 69, 93, 및 115에 제시된다. 항체의 VH CDR2의 아미노산 서열은 서열 70, 94, 및 116에 제시된다. 항체의 VH CDR3의 아미노산 서열은 서열 71, 95, 및 117에 제시된다. 항체의 VL CDR1의 아미노산 서열은 서열 72, 96, 및 118에 제시된다. 항체의 VL CDR2의 아미노산 서열은 서열 73, 97, 및 119에 제시된다. 항체의 VL CDR3의 아미노산 서열은 서열 74, 98, 및 120에 제시된다. CDR 영역은 카바트 시스템을 사용하여 기재된다 ([Kabat et al., (1991) Sequences of Proteins of Immunological Interest, Fifth Edition, U.S. Department of Health and Human Services, NIH Publication No. 91-3242]; [Chothia et al., (1987) J. Mol. Biol. 196: 901-917]; [Chothia et al., (1989) Nature 342: 877-883]; 및 [Al-Lazikani et al., (1997) J. Mol. Biol. 273, 927-948]).
각각의 이들 항체가 LRP6에 결합할 수 있고 항원-결합 특이성이 주로 CDR1, 2 및 3 영역에 의해 제공됨을 고려하면, VH CDR1, 2 및 3 서열 및 VL CDR1, 2 및 3 서열은 "혼합 및 매치"될 수 있지만 (즉, 상이한 항체로부터의 CDR이 혼합 및 매치될 수 있음), 각각의 항체는 본 발명의 다른 LRP6을 생성하기 위해 VH CDR1, 2 및 3 및 VL CDR1, 2 및 3을 함유해야 한다. 이러한 "혼합 및 매치"된 LRP6 항체는 당업계에 공지된 결합 검정 및 실시예에 기재된 방법 (예를 들어, ELISA)으로 시험할 수 있다. VH CDR 서열이 혼합 및 매치되는 경우, 특정 VH 서열로부터의 CDR1, CDR2 및/또는 CDR3 서열은 구조적으로 유사한 CDR 서열(들)로 대체되어야 한다. 마찬가지로, VL CDR 서열이 혼합 및 매치되는 경우, 특정 VL 서열로부터의 CDR1, CDR2 및/또는 CDR3 서열은 구조적으로 유사한 CDR 서열(들)로 대체되어야 한다. 1개 이상의 VH 및/또는 VL CDR 영역 서열을 본 발명의 모노클로날 항체에 대해 본원에 나타낸 CDR 서열로부터의 구조적으로 유사한 서열로 치환함으로써 신규한 VH 및 VL 서열이 생성될 수 있음이 당업자에게 매우 명확할 것이다.
따라서, 본 발명은 서열 1, 21, 및 47로 이루어진 군 중에서 선택된 아미노산 서열을 포함하는 중쇄 가변 영역 CDR1; 서열 2, 22, 및 48로 이루어진 군 중에서 선택된 아미노산 서열을 포함하는 중쇄 가변 영역 CDR2; 서열 3, 23, 및 49로 이루어진 군 중에서 선택된 아미노산 서열을 포함하는 중쇄 가변 영역 CDR3; 서열 4, 24, 및 50으로 이루어진 군 중에서 선택된 아미노산 서열을 포함하는 경쇄 가변 영역 CDR1; 서열 5, 25, 및 51로 이루어진 군 중에서 선택된 아미노산 서열을 포함하는 경쇄 가변 영역 CDR2; 및 서열 6, 26, 및 52로 이루어진 군 중에서 선택된 아미노산 서열을 포함하는 경쇄 가변 영역 CDR3을 포함하는, LRP6에 결합하는 단리된 LRP6 β-프로펠러 1 모노클로날 항체 또는 그의 단편을 제공한다.
따라서, 본 발명은 서열 69, 93, 및 115로 이루어진 군 중에서 선택된 아미노산 서열을 포함하는 중쇄 가변 영역 CDR1; 서열 70, 94, 및 116으로 이루어진 군 중에서 선택된 아미노산 서열을 포함하는 중쇄 가변 영역 CDR2; 서열 71, 95, 및 117로 이루어진 군 중에서 선택된 아미노산 서열을 포함하는 중쇄 가변 영역 CDR3; 서열 72, 96, 및 118로 이루어진 군 중에서 선택된 아미노산 서열을 포함하는 경쇄 가변 영역 CDR1; 서열 73, 97, 및 119로 이루어진 군 중에서 선택된 아미노산 서열을 포함하는 경쇄 가변 영역 CDR2; 및 서열 74, 98, 및 120으로 이루어진 군 중에서 선택된 아미노산 서열을 포함하는 경쇄 가변 영역 CDR3을 포함하는, LRP6에 결합하는 단리된 LRP6 β-프로펠러 3 모노클로날 항체 또는 그의 단편을 제공한다.
특정 실시양태에서, LRP6에 결합하는 항체는 서열 1의 중쇄 가변 영역 CDR1; 서열 2의 중쇄 가변 영역 CDR2; 서열 3의 중쇄 가변 영역 CDR3; 서열 4의 경쇄 가변 영역 CDR1; 서열 5의 경쇄 가변 영역 CDR2; 및 서열 6의 경쇄 가변 영역 CDR3을 포함한다.
특정 실시양태에서, LRP6에 결합하는 항체는 서열 21의 중쇄 가변 영역 CDR1; 서열 22의 중쇄 가변 영역 CDR2; 서열 23의 중쇄 가변 영역 CDR3; 서열 24의 경쇄 가변 영역 CDR1; 서열 25의 경쇄 가변 영역 CDR2; 및 서열 26의 경쇄 가변 영역 CDR3을 포함한다.
특정 실시양태에서, LRP6에 결합하는 항체는 서열 47의 중쇄 가변 영역 CDR1; 서열 48의 중쇄 가변 영역 CDR2; 서열 49의 중쇄 가변 영역 CDR3; 서열 50의 경쇄 가변 영역 CDR1; 서열 51의 경쇄 가변 영역 CDR2; 및 서열 52의 경쇄 가변 영역 CDR3을 포함한다.
특정 실시양태에서, LRP6에 결합하는 항체는 서열 69의 중쇄 가변 영역 CDR1; 서열 70의 중쇄 가변 영역 CDR2; 서열 71의 중쇄 가변 영역 CDR3; 서열 72의 경쇄 가변 영역 CDR1; 서열 73의 경쇄 가변 영역 CDR2; 및 서열 74의 경쇄 가변 영역 CDR3을 포함한다.
특정 실시양태에서, LRP6에 결합하는 항체는 서열 93의 중쇄 가변 영역 CDR1; 서열 94의 중쇄 가변 영역 CDR2; 서열 95의 중쇄 가변 영역 CDR3; 서열 96의 경쇄 가변 영역 CDR1; 서열 97의 경쇄 가변 영역 CDR2; 및 서열 98의 경쇄 가변 영역 CDR3을 포함한다.
특정 실시양태에서, LRP6에 결합하는 항체는 서열 115의 중쇄 가변 영역 CDR1; 서열 116의 중쇄 가변 영역 CDR2; 서열 117의 중쇄 가변 영역 CDR3; 서열 118의 경쇄 가변 영역 CDR1; 서열 119의 경쇄 가변 영역 CDR2; 및 서열 120의 경쇄 가변 영역 CDR3을 포함한다.
특정 실시양태에서, LRP6에 결합하는 항체는 서열 14의 VH 및 서열 13의 VL을 포함한다. 특정 실시양태에서, LRP6에 결합하는 항체는 서열 34의 VH 및 서열 33의 VL을 포함한다. 특정 실시양태에서, LRP6에 결합하는 항체는 서열 35의 VH 및 서열 36의 VL을 포함한다. 특정 실시양태에서, LRP6에 결합하는 항체는 서열 43의 VH 및 서열 44의 VL을 포함한다. 특정 실시양태에서, LRP6에 결합하는 항체는 서열 60의 VH 및 서열 59의 VL을 포함한다. 특정 실시양태에서, LRP6에 결합하는 항체는 서열 62의 VH 및 서열 61의 VL을 포함한다. 특정 실시양태에서, LRP6에 결합하는 항체는 서열 82의 VH 및 서열 81의 VL을 포함한다. 특정 실시양태에서, LRP6에 결합하는 항체는 서열 90의 VH 및 서열 89의 VL을 포함한다. 특정 실시양태에서, LRP6에 결합하는 항체는 서열 106의 VH 및 서열 105의 VL을 포함한다. 특정 실시양태에서, LRP6에 결합하는 항체는 서열 108의 VH 및 서열 107의 VL을 포함한다. 특정 실시양태에서, LRP6에 결합하는 항체는 서열 128의 VH 및 서열 127의 VL을 포함한다. 특정 실시양태에서, LRP6에 결합하는 항체는 서열 130의 VH 및 서열 129의 VL을 포함한다. 특정 실시양태에서, LRP6에 결합하는 항체는 서열 138의 VH 및 서열 137의 VL을 포함한다.
한 실시양태에서, LRP6 항체는 길항제 항체이다. 한 실시양태에서, LRP6 항체는 효능제 항체이다. 특정 실시양태에서, LRP6에 결합하는 항체는 표 1에 기재된 항체이다.
본원에서 사용된 바와 같이, 인간 항체는 항체의 가변 영역 또는 전장 쇄가 인간 배선 이뮤노글로불린 유전자를 사용한 시스템으로부터 수득된 경우에 특정 배선 서열"의 생성물"이거나 "그로부터 유도된" 중쇄 또는 경쇄 가변 영역 또는 전장 중쇄 또는 경쇄를 포함한다. 이러한 시스템은 인간 이뮤노글로불린 유전자를 보유하는 트랜스제닉 마우스를 관심있는 항원으로 면역화시키거나, 또는 파지에 디스플레이된 인간 이뮤노글로불린 유전자 라이브러리를 관심있는 항원을 사용하여 스크리닝하는 것을 포함한다. 인간 배선 이뮤노글로불린 서열"의 생성물"이거나 "그로부터 유도된" 인간 항체는 인간 항체의 아미노산 서열을 인간 배선 이뮤노글로불린의 아미노산 서열과 비교하고, 인간 항체 서열에 가장 근접한 서열 (즉, 최대 % 동일성)인 인간 배선 이뮤노글로불린 서열을 선택함으로써 확인할 수 있다. 특정 인간 배선 이뮤노글로불린 서열"의 생성물"이거나 "그로부터 유도된" 인간 항체는 배선 서열에 비해, 예를 들어 천연 생성 체세포 돌연변이, 또는 부위-지정 돌연변이의 의도적인 도입으로 인한 아미노산 차이를 가질 수 있다. 그러나, VH 또는 VL 프레임워크 영역에서, 선택된 인간 항체는 일반적으로 인간 배선 이뮤노글로불린 유전자에 의해 코딩되는 아미노산 서열에 대한 아미노산 서열 동일성이 적어도 90%이고, 다른 종의 배선 이뮤노글로불린 아미노산 서열 (예컨대, 뮤린 배선 서열)과 비교할 때 인간 항체를 인간의 것으로 확인시켜 주는 아미노산 잔기를 함유한다. 특정한 경우, 인간 항체는 배선 이뮤노글로불린 유전자에 의해 코딩되는 아미노산 서열에 대한 아미노산 서열 동일성이 적어도 60%, 70%, 80%, 90% 또는 적어도 95% 또는 심지어는 적어도 96%, 97%, 98% 또는 99%일 수 있다. 일반적으로, 재조합 인간 항체는 VH 또는 VL 프레임워크 영역에서 인간 배선 이뮤노글로불린 유전자에 의해 코딩되는 아미노산 서열과 10개 이하의 아미노산 차이를 보일 것이다. 특정한 경우, 인간 항체는 배선 이뮤노글로불린 유전자에 의해 코딩되는 아미노산 서열과 5개 이하, 또는 심지어는 4개, 3개, 2개 또는 1개 이하의 아미노산 차이를 나타낼 수 있다.
본원에 개시되는 항체는 단일 쇄 항체, 디아바디, 도메인 항체, 나노바디 (nanobody), 및 유니바디의 유도체일 수 있다. "단일 쇄 항체" (scFv)는 VH 도메인에 연결된 VL 도메인을 포함하는 단일 폴리펩티드 사슬로 이루어지고, 여기서, VL 도메인 및 VH 도메인은 페어링하여 일가 분자를 형성한다. 단일 쇄 항체는 당업계에 공지된 방법에 따라 제조될 수 있다 (예를 들어, 문헌 [Bird et al., (1988) Science 242:423-426] 및 [Huston et al., (1988) Proc. Natl. Acad. Sci. USA 85:5879-5883] 참조). "디스버드(disbud)"는 짧은 펩티드 링커에 의해 연결된 동일한 폴리펩티드 사슬 상의 경쇄 가변 영역에 연결된 중쇄 가변 영역을 각각 포함하는 2개의 사슬로 이루어지고, 여기서, 동일한 사슬 상의 2개의 영역은 서로 페어링하지 않지만 다른 사슬 상의 상보성 도메인과는 페어링하여 이중특이적 분자를 형성한다. 디아바디의 제조 방법은 당업계에 공지되어 있다 (예를 들어, 문헌 [Holliger et al., (1993) Proc. Natl. Acad. Sci. USA 90:6444-6448], 및 [Poljak et al., (1994) Structure 2:1121-1123]). 도메인 항체 (dAb)는 항체의 중쇄 또는 경쇄의 가변 영역에 대응하는 항체의 작은 기능적 결합 단위이다. 도메인 항체는 박테리아, 효모, 및 포유동물 세포 시스템에서 잘 발현된다. 도메인 항체 및 그의 생산 방법의 추가의 상세한 내용은 당업계에 공지되어 있다 (예를 들어, 미국 특허 번호 6,291,158; 6,582,915; 6,593,081; 6,172,197; 6,696,245; 유럽 특허 0368684 & 0616640; WO05/035572, WO04/101790, WO04/081026, WO04/058821, WO04/003019 및 WO03/002609 참조). 나노바디는 항체의 중쇄로부터 유래한다. 나노바디는 일반적으로 단일 가변 도메인 및 2개의 불변 도메인 (CH2 및 CH3)을 포함하고, 본래의 항체의 항원-결합 능력을 보유한다. 나노바디는 당업계에 공지된 방법에 의해 제조할 수 있다 (예를 들어, 미국 특허 번호 6,765,087, 미국 특허 번호 6,838,254, WO 06/079372 참조). 유니바디는 IgG4 항체의 하나의 경쇄 및 하나의 중쇄로 이루어진다. 유니바디는 IgG4 항체의 힌지 영역의 제거에 의해 제조될 수 있다. 유니바디 및 그의 제조 방법의 추가의 상세한 내용은 WO2007/059782에서 찾을 수 있다.
Wnt 리간드에 추가로, LRP6 프로펠러 1 항체는 다른 프로펠러 1 결합 리간드 (예를 들어 스클레로스틴, Dkk1)와의 상호작용을 억제할 것으로 예상된다. 이와 유사하게, 프로펠러 3 항체는 다른 프로펠러 3 결합 리간드 (예를 들어 Dkk1)와의 상호작용을 억제할 것으로 예상된다. 또한, 프로펠러 1 및 3 결합 항체는 다른 Wnt 신호 전달 조절물질, 예를 들어 R-스폰딘의 활성에 영향을 줄 것으로 예상될 수 있다.
상동성
항체
또 다른 실시양태에서, 본 발명은 표 1에 기재된 서열에 상동성인 아미노산 서열을 포함하는 항체 또는 그의 단편을 제공하고, 항체는 LRP6 단백질 (예를 들어, 인간 및/또는 시노몰구스 LRP6)에 결합하고, 표 1에 기재된 항체의 목적하는 기능적 특성을 보유한다.
예를 들어, 본 발명은 서열 14, 34, 36, 44, 60, 및 62로 이루어진 군 중에서 선택된 아미노산 서열과 적어도 80%, 적어도 90% 또는 적어도 95% 동일한 아미노산 서열을 포함하는 중쇄 가변 영역, 및 서열 13, 33, 37, 43, 59, 및 61로 이루어지는 군 중에서 선택된 아미노산 서열과 적어도 80%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일한 아미노산 서열을 포함하는 경쇄 가변 영역을 포함하고, LRP6 (예를 들어, 인간 및/또는 시노몰구스 LRP6)의 β-프로펠러 1에 결합하고, 본원에서 설명되는 Wnt 리포터 유전자 검정 또는 Wnt 지정 신호 전달의 다른 척도 (예를 들어, LRP6 인산화, β-카테닌 안정화 및 핵 전위, 세포 증식/생존)에서 측정될 수 있는 바와 같이 β-프로펠러 1 의존 Wnt 단백질의 신호 전달 활성을 억제하는 단리된 모노클로날 항체 (또는 그의 기능적 단편)를 제공한다. 구체적인 예에서, 상기 항체의 Wnt1 검정에서의 EC50 값은 조건화 배지 또는 형질감염된 세포를 사용할 때 10 nM 미만이다.
예를 들어, 본 발명은 서열 82, 89, 106, 108, 128, 130, 및 138로 이루어진 군 중에서 선택된 아미노산 서열과 적어도 80%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일한 아미노산 서열을 포함하는 중쇄 가변 영역, 및 서열 81, 90, 105, 107, 127, 129, 및 137로 이루어지는 군 중에서 선택된 아미노산 서열과 적어도 80%, 적어도 90%, 적어도 95%, 또는 적어도 98% 동일한 아미노산 서열을 포함하는 경쇄 가변 영역을 포함하고, LRP6 (예를 들어, 인간 및/또는 시노몰구스 LRP6)의 β-프로펠러 3에 결합하고, 본원에서 설명되는 Wnt 리포터 유전자 검정 또는 Wnt 지정 신호 전달의 다른 척도 (예를 들어, LRP6 인산화, β-카테닌 안정화 및 핵 전위, 세포 증식/생존)에서 측정될 수 있는 바와 같이 β-프로펠러 3 의존 Wnt 단백질의 신호 전달 활성을 억제하는 단리된 모노클로날 항체 (또는 그의 기능적 단편)를 제공한다. 구체적인 예에서, 상기 항체의 Wnt3a 검정에서의 EC50 값은 조건화 배지 또는 형질감염된 세포를 사용할 때 10 nM 미만이다.
추가로, 프로펠러 1 항체의 경우, 가변 중쇄 모 뉴클레오티드 서열은 서열 16, 38, 및 64에 제시된다. 가변 경쇄 모 뉴클레오티드 서열은 서열 15, 37, 및 63에 제시된다. 포유동물 세포에서의 발현에 최적화된 전장 중쇄 서열은 서열 20, 42 및 68에 제시된다. 포유동물 세포에서의 발현에 최적화된 전장 경쇄 서열은 서열 19, 41 및 67에 제시된다. 본 발명의 다른 항체는, 돌연변이되었지만 상기한 서열에 대해 적어도 60%, 70%, 80%, 90%, 95% 또는 98% 동일성을 갖는 아미노산 또는 핵산을 포함한다. 몇몇 실시양태에서, 항체는 1, 2, 3, 4 또는 5개 이하의 아미노산이 상기한 서열에 제시된 가변 영역과 비교할 때 가변 영역 내의 아미노산 결실, 삽입 또는 치환에 의해 돌연변이된 돌연변이체 아미노산 서열을 포함한다.
추가로, 프로펠러 3 항체의 경우, 가변 중쇄 모 뉴클레오티드 서열은 서열 84, 110, 및 132에 제시된다. 가변 경쇄 모 뉴클레오티드 서열은 서열 83, 109, 및 131에 제시된다. 포유동물 세포에서의 발현에 최적화된 전장 중쇄 서열은 서열 88, 91, 114, 136, 및 140에 제시된다. 포유동물 세포에서의 발현에 최적화된 전장 경쇄 서열은 서열 87, 92, 113, 135, 및 139에 제시된다. 본 발명의 다른 항체는, 돌연변이되었지만 상기한 서열에 대해 적어도 60%, 70%, 80%, 90%, 95% 또는 98% 동일성을 갖는 아미노산 또는 핵산을 포함한다. 몇몇 실시양태에서, 항체는 1, 2, 3, 4 또는 5개 이하의 아미노산이 상기한 서열에 제시된 가변 영역과 비교할 때 가변 영역 내의 아미노산 결실, 삽입 또는 치환에 의해 돌연변이된 돌연변이체 아미노산 서열을 포함한다.
다른 실시양태에서, VH 및/또는 VL 아미노산 서열은 표 1에 제시된 서열에 50%, 60%, 70%, 80%, 90%, 95%, 96%, 97%, 98% 또는 99% 동일할 수 있다. 다른 실시양태에서, VH 및/또는 VL 아미노산 서열은 1, 2, 3, 4 또는 5개 이하의 아미노산 위치에서의 아미노산 치환을 제외하고 동일할 수 있다. 표 1에 기재된 프로펠러 1 항체의 VH 및 VL 영역에 높은 (즉, 80% 이상의) 동일성을 갖는 VH 및 VL 영역을 갖는 항체는 서열 14, 34, 60, 13, 33, 및 59를 각각 코딩하는 핵산 분자의 돌연변이 유발 (예를 들어, 부위-지정 또는 PCR-매개 돌연변이 유발) 및 이후 상기 코딩된 변경된 항체를 본원에 기재된 기능성 검정을 이용하여 보유된 기능에 대해 시험함으로써 수득될 수 있다.
표 1에 기재된 프로펠러 3 항체의 VH 및 VL 영역에 높은 (즉, 80% 이상의) 동일성을 갖는 VH 및 VL 영역을 갖는 항체는 서열 82, 106, 128, 81, 105, 및 127을 각각 코딩하는 핵산 분자의 돌연변이 유발 (예를 들어, 부위-지정 또는 PCR-매개 돌연변이 유발) 및 이후 상기 코딩된 변경된 항체를 본원에 기재된 기능성 검정을 이용하여 보유된 기능에 대해 시험함으로써 수득될 수 있다.
다른 실시양태에서, 중쇄 및/또는 경쇄 뉴클레오티드 서열의 가변 영역은 상기 제시된 서열에 60%, 70%, 80%, 90%, 95%, 96%, 97%, 98% 또는 99% 동일할 수 있다.
본원에서 사용되는 바와 같이, 2개의 서열들 사이의 "동일성 %"는 2개 서열의 최적 정렬을 위해 도입될 필요가 있는 갭의 수 및 각각의 갭의 길이를 고려하여, 서열들에 의해 공유되는 동일한 위치의 수의 함수이다 (즉, 동일성 % = (동일한 위치의 수/위치의 총 수) x 100). 2개의 서열 사이의 서열 비교 및 동일성 %의 결정은 아래의 비-제한적인 예에 기재된 바와 같이 수학적 알고리즘을 이용하여 이루어질 수 있다.
추가로 또는 별법으로, 본 발명의 단백질 서열은 예를 들어 관련 서열들을 확인하기 위해 공공 데이터베이스에 대한 검색을 수행하기 위한 "질의 서열"로서 추가로 사용될 수도 있다. 예를 들어, 이러한 검색은 문헌 [Altschul, et al., (1990) J. Mol. Biol. 215:403-10]의 BLAST 프로그램 (버전 2.0)을 이용하여 수행할 수 있다.
보존적 변형을 갖는 항체
특정 실시양태에서, 본 발명의 항체는 CDR1, CDR2 및 CDR3 서열을 포함하는 중쇄 가변 영역, 및 CDR1, CDR2 및 CDR3 서열을 포함하는 경쇄 가변 영역을 갖고, 여기서 이들 CDR 서열 중 1개 이상은 본원에 기재된 항체 또는 그의 보존적 변형을 기초로 하는 특정 아미노산 서열을 갖고, 상기 항체는 본 발명의 LRP6 항체의 목적하는 기능적 특성을 보유한다.
따라서, 본 발명은 CDR1, CDR2 및 CDR3 서열을 포함하는 중쇄 가변 영역, 및 CDR1, CDR2 및 CDR3 서열을 포함하는 경쇄 가변 영역으로 이루어지는 단리된 프로펠러 1 모노클로날 항체, 또는 그의 기능적 단편을 제공하고, 여기서, 중쇄 가변 영역의 CDR1 아미노산 서열은 서열 1, 21, 및, 47, 및 그의 보존적 변형으로 이루어지는 군 중에서 선택되고; 중쇄 가변 영역의 CDR2 아미노산 서열은 서열 2, 22, 및 48, 및 그의 보존적 변형으로 이루어지는 군 중에서 선택되고; 중쇄 가변 영역의 CDR3 아미노산 서열은 서열 3, 23, 및 49, 및 그의 보존적 변형으로 이루어지는 군 중에서 선택되고; 경쇄 가변 영역의 CDR1 아미노산 서열은 서열 4, 24, 50, 및 그의 보존적 변형으로 이루어지는 군 중에서 선택되고; 경쇄 가변 영역의 CDR2 아미노산 서열은 서열 5, 25, 및 51, 및 그의 보존적 변형으로 이루어지는 군 중에서 선택되고; 경쇄 가변 영역의 CDR3 아미노산 서열은 서열 6, 26, 및 52, 및 그의 보존적 변형으로 이루어지는 군 중에서 선택되고; 항체 또는 그의 단편은 LRP6에 특이적으로 결합하고, Wnt 신호 전달 경로를 억제함으로써 LRP6 활성을 억제하고, 이는 본원에서 설명되는 바와 같은 Wnt 리포터 유전자 검정 또는 Wnt 지정 신호 전달의 다른 척도 (예를 들어, LRP6 인산화, β-카테닌 안정화 및 핵 전위, 세포 증식/생존)에서 측정될 수 있다.
따라서, 본 발명은 CDR1, CDR2 및 CDR3 서열을 포함하는 중쇄 가변 영역, 및 CDR1, CDR2 및 CDR3 서열을 포함하는 경쇄 가변 영역으로 이루어지는 단리된 프로펠러 3 모노클로날 항체, 또는 그의 단편을 제공하고, 여기서, 중쇄 가변 영역의 CDR1 아미노산 서열은 서열 69, 93, 및 115, 및 그의 보존적 변형으로 이루어지는 군 중에서 선택되고; 중쇄 가변 영역의 CDR2 아미노산 서열은 서열 70, 94, 및 116, 및 그의 보존적 변형으로 이루어지는 군 중에서 선택되고; 중쇄 가변 영역의 CDR3 아미노산 서열은 서열 71, 95, 및 117, 및 그의 보존적 변형으로 이루어지는 군 중에서 선택되고; 경쇄 가변 영역의 CDR1 아미노산 서열은 서열 72, 96, 및 118, 및 그의 보존적 변형으로 이루어지는 군 중에서 선택되고; 경쇄 가변 영역의 CDR2 아미노산 서열은 서열 73, 97, 및 119, 및 그의 보존적 변형으로 이루어지는 군 중에서 선택되고; 경쇄 가변 영역의 CDR3 아미노산 서열은 서열 74, 98, 및 120, 및 그의 보존적 변형으로 이루어지는 군 중에서 선택되고; 항체 또는 그의 단편은 LRP6에 특이적으로 결합하고, 프로펠러 3 의존 Wnt 단백질의 활성을 억제하고, 이것은 본원에서 설명되는 바와 같은 Wnt 리포터 유전자 검정 또는 Wnt 지정 신호 전달의 다른 척도 (예를 들어, LRP6 인산화, β-카테닌 안정화 및 핵 전위, 세포 증식/생존)에서 측정될 수 있다.
동일한
에피토프에
결합하는 항체
본 발명은 표 1에 기재된 LRP6 항체와 동일한 에피토프에 결합하는 항체를 제공한다. 따라서, 추가의 항체는 LRP6 결합 검정에서 본 발명의 다른 항체와 교차-경쟁하는 (예를 들어, 본 발명의 다른 항체의 결합을 통계적으로 유의한 방식으로 경쟁적으로 억제하는) 능력을 기초로 하여 확인될 수 있다. LRP6 단백질 (예를 들어, 인간 및/또는 시노몰구스 LRP6)에 대한 본 발명의 항체의 결합을 억제하는 시험 항체의 능력은 시험 항체가 LRP6에 대한 결합을 위해 상기 항체와 경쟁할 수 있음을 입증하고, 이러한 항체는 비-제한적인 이론에 따라 이것이 경쟁하는 항체와 동일하거나 관련이 있는 (예를 들어, 구조적으로 유사하거나 공간적으로 근접한), LRP6 단백질 상의 에피토프에 결합할 수 있다. 특정 실시양태에서, 본 발명의 항체와 LRP6의 동일 에피토프에 결합하는 항체는 인간 모노클로날 항체이다. 이러한 인간 모노클로날 항체는 본원에 기재된 바와 같이 제조 및 단리될 수 있다.
조작 및 변형된 항체
추가로, 본 발명의 항체는, 변형된 항체를 조작하기 위한 출발 물질로서 본원에 제시된 1종 이상의 VH 및/또는 VL 서열을 갖는 항체를 사용하여 제조될 수 있고, 상기 변형된 항체는 출발 항체로부터 변경된 특성을 가질 수 있다. 항체는 1개 또는 2개 모두의 가변 영역 (즉, VH 및/또는 VL) 내에, 예를 들어 1개 이상의 CDR 영역 내에 및/또는 1개 이상의 프레임워크 영역 내에 존재하는 1개 이상의 잔기를 변형시켜 조작될 수 있다. 추가로 또는 별법으로, 항체는 불변 영역(들) 내의 잔기들을 변형시켜 조작되어, 예를 들어 항체의 이펙터 기능(들)을 변경시킬 수 있다.
수행될 수 있는 가변 영역 조작의 유형 중 하나는 CDR 그라프팅이다. 항체는 주로 6개의 중쇄 및 경쇄 상보성 결정 영역 (CDR) 내에 위치하는 아미노산 잔기를 통해 표적 항원과 상호작용한다. 이러한 이유로 인해, CDR 내의 아미노산 서열은 CDR 외부의 서열보다 개개의 항체들 사이에서 더 다양하다. CDR 서열이 대부분의 항체-항원 상호작용을 담당하기 때문에, 상이한 특성을 갖는 상이한 항체로부터의 프레임워크 서열에 그라프팅된, 특정 천연 생성 항체로부터의 CDR 서열을 포함하는 발현 벡터를 제작함으로써 특정 천연 생성 항체의 특성을 모방하는 재조합 항체를 발현시키는 것이 가능하다 (예를 들어, 문헌 [Riechmann et al., (1998) Nature 332:323-327]; [Jones et al., (1986) Nature 321:522-525]; [Queen et al., (1989) Proc. Natl. Acad., U.S.A. 86:10029-10033]; 미국 특허 번호 5,225,539 (윈터(Winter)), 및 미국 특허 번호 5,530,101; 5,585,089; 5,693,762 및 6,180,370 (퀸(Queen) 등) 참조)
따라서, 본 발명의 또 다른 실시양태는 서열 1, 21, 및 47로 이루어진 군 중에서 선택된 아미노산 서열을 갖는 CDR1 서열; 서열 2, 22, 및 48로 이루어진 군 중에서 선택된 아미노산 서열을 갖는 CDR2 서열; 서열 3, 23, 및 49로 이루어진 군 중에서 선택된 아미노산 서열을 갖는 CDR3 서열을 포함하는 중쇄 가변 영역; 및 서열 4, 24, 및 50으로 이루어진 군 중에서 선택된 아미노산 서열을 갖는 CDR1 서열; 서열 5, 25, 및 51로 이루어진 군 중에서 선택된 아미노산 서열을 갖는 CDR2 서열; 및 서열 6, 26, 및 52로 이루어진 군 중에서 선택된 아미노산 서열로 이루어지는 CDR3 서열을 갖는 경쇄 가변 영역을 포함하는, 단리된 프로펠러 1 모노클로날 항체 또는 그의 단편에 관한 것이다. 따라서, 상기 항체는 모노클로날 항체의 VH 및 VL CDR 서열을 함유하지만, 이들 항체의 상이한 프레임워크 서열을 함유할 수 있다.
따라서, 본 발명의 또 다른 실시양태는 서열 69, 93, 및 115로 이루어진 군 중에서 선택된 아미노산 서열을 갖는 CDR1 서열; 서열 70, 100, 및 116으로 이루어진 군 중에서 선택된 아미노산 서열을 갖는 CDR2 서열; 서열 71, 95, 및 117로 이루어진 군 중에서 선택된 아미노산 서열을 갖는 CDR3 서열을 포함하는 중쇄 가변 영역; 및 서열 72, 96, 및 118로 이루어진 군 중에서 선택된 아미노산 서열을 갖는 CDR1 서열; 서열 73, 97, 및 119로 이루어진 군 중에서 선택된 아미노산 서열을 갖는 CDR2 서열; 및 서열 74, 98, 및 120으로 이루어진 군 중에서 선택된 아미노산 서열로 이루어지는 CDR3 서열을 갖는 경쇄 가변 영역을 포함하는, 단리된 프로펠러 3 모노클로날 항체 또는 그의 단편에 관한 것이다. 따라서, 상기 항체는 모노클로날 항체의 VH 및 VL CDR 서열을 함유하지만, 이들 항체의 상이한 프레임워크 서열을 함유할 수 있다.
이러한 프레임워크 서열은 배선 항체 유전자 서열을 포함하는 공공 DNA 데이터베이스 또는 간행된 문헌으로부터 수득할 수 있다. 예를 들어, 인간 중쇄 및 경쇄 가변 영역 유전자에 대한 배선 DNA 서열은 "Vase" 인간 배선 서열 데이터베이스 (인터넷 웹사이트 www.mrc-cpe.cam.ac.uk/vbase에서 이용가능함), 및 또한 문헌 [Kabat et al., (1991) Sequences of Proteins of Immunological Interest, Fifth Edition, U.S. Department of Health and Human Services, NIH Publication No. 91-3242], [Chothia et al., (1987) J. Mol. Biol. 196:901-917]; [Chothia et al., (1989) Nature 342:877-883]; 및 [Al-Lazikani et al., (1997) J. Mol. Biol. 273:927-948]; [Tomlinson et al., (1992) J. fol. Biol. 227:776-798] 및 [Cox et al., (1994) Eur. J Immunol. 24:827-836] (이들 문헌의 각각의 내용은 본원에 명백하게 참고로 포함됨)에서 찾을 수 있다.
본 발명의 항체에 사용하기 위한 프레임워크 서열의 예는 본 발명의 선택된 항체에 의해 사용되는 프레임워크 서열, 예를 들어 본 발명의 모노클로날 항체에 의해 사용되는 컨센서스 서열 및/또는 프레임워크 서열에 구조적으로 유사한 것이다. VH CDR1, 2 및 3 서열, 및 VL CDR1, 2 및 3 서열을 프레임워크 서열이 유래되는 배선 이뮤노글로불린 유전자에서 발견되는 것과 동일한 서열을 갖는 프레임워크 영역으로 그라프팅시킬 수 있거나, 또는 CDR 서열을 배선 서열과 비교해서 1개 이상의 돌연변이를 함유하는 프레임워크 영역으로 그라프팅시킬 수도 있다. 예를 들어, 특정 예에서는 프레임워크 영역 내의 잔기를 돌연변이시켜 항체의 항원 결합 능력을 유지 또는 증진시키는 것이 유익한 것으로 밝혀졌다 (예를 들어, 미국 특허 번호 5,530,101; 5,585,089; 5,693,762 및 6,180,370 (퀸 등) 참조).
또 다른 유형의 가변 영역 변형은 VH 및/또는 VL CDR1, CDR2 및/또는 CDR3 영역 내의 아미노산 잔기를 돌연변이시켜 관심있는 항체의 하나 이상의 결합 특성 (예를 들어, 친화도)을 개선하는 것이다 ("친화도 성숙"으로 공지됨). 부위-지정 돌연변이 유발 또는 PCR-매개 돌연변이 유발을 수행하여 돌연변이(들)을 도입할 수 있고, 항체 결합에 대한 효과 또는 관심의 대상이 되는 다른 기능적 특성을 본원에 기재되고 실시예에서 제시되는 바와 같은 시험관내 또는 생체내 검정으로 평가할 수 있다. 보존적 변형 (상기 논의된 바와 같음)이 도입될 수 있다. 돌연변이는 아미노산 치환, 부가 또는 결실일 수 있다. 추가로, 일반적으로는 CDR 영역 내의 1개, 2개, 3개, 4개 또는 5개 이하의 잔기를 변경시킨다.
따라서, 또 다른 실시양태에서, 본 발명은 서열 1, 21, 및 47을 갖는 군 중에서 선택된 아미노산 서열 또는 서열 1, 21, 및 47과 비교하여 1개, 2개, 3개, 4개 또는 5개의 아미노산 치환, 결실 또는 부가를 갖는 아미노산 서열로 이루어진 VH CDR1 영역; 서열 2, 22, 및 48로 이루어진 군 중에서 선택된 아미노산 서열 또는 서열 2, 22, 및 48과 비교하여 1개, 2개, 3개, 4개 또는 5개의 아미노산 치환, 결실 또는 부가를 갖는 아미노산 서열을 갖는 VH CDR2 영역; 서열 3, 23, 및 49로 이루어진 군 중에서 선택된 아미노산 서열 또는 서열 3, 23, 및 49와 비교하여 1개, 2개, 3개, 4개 또는 5개의 아미노산 치환, 결실 또는 부가를 갖는 아미노산 서열을 갖는 VH CDR3 영역을 갖는 중쇄 가변 영역; 서열 4, 24, 및 50으로 이루어진 군 중에서 선택된 아미노산 서열 또는 서열 4, 24, 및 50과 비교하여 1개, 2개, 3개, 4개 또는 5개의 아미노산 치환, 결실 또는 부가를 갖는 아미노산 서열을 갖는 VL CDR1 영역; 서열 5, 25, 및 51로 이루어진 군 중에서 선택된 아미노산 서열 또는 서열 5, 25, 및 51과 비교하여 1개, 2개, 3개, 4개 또는 5개의 아미노산 치환, 결실 또는 부가를 갖는 아미노산 서열을 갖는 VL CDR2 영역; 및 서열 6, 26, 및 52로 이루어진 군 중에서 선택된 아미노산 서열 또는 서열 6, 26, 및 52와 비교하여 1개, 2개, 3개, 4개 또는 5개의 아미노산 치환, 결실 또는 부가를 갖는 아미노산 서열을 갖는 VL CDR3 영역으로 이루어진 단리된 프로펠러 1 모노클로날 항체, 또는 그의 단편을 제공한다.
따라서, 또 다른 실시양태에서, 본 발명은 서열 69, 93, 및 115를 갖는 군 중에서 선택된 아미노산 서열 또는 서열 69, 93, 및 115와 비교하여 1개, 2개, 3개, 4개 또는 5개의 아미노산 치환, 결실 또는 부가를 갖는 아미노산 서열로 이루어진 VH CDR1 영역; 서열 70, 94, 및 116으로 이루어진 군 중에서 선택된 아미노산 서열 또는 서열 70, 94, 및 116과 비교하여 1개, 2개, 3개, 4개 또는 5개의 아미노산 치환, 결실 또는 부가를 갖는 아미노산 서열을 갖는 VH CDR2 영역; 서열 71, 95, 및 117로 이루어진 군 중에서 선택된 아미노산 서열 또는 서열 71, 95, 및 117과 비교하여 1개, 2개, 3개, 4개 또는 5개의 아미노산 치환, 결실 또는 부가를 갖는 아미노산 서열을 갖는 VH CDR3 영역을 갖는 중쇄 가변 영역; 서열 72, 96, 및 118로 이루어진 군 중에서 선택된 아미노산 서열 또는 서열 72, 96, 및 118과 비교하여 1개, 2개, 3개, 4개 또는 5개의 아미노산 치환, 결실 또는 부가를 갖는 아미노산 서열을 갖는 VL CDR1 영역; 서열 73, 97, 및 119로 이루어진 군 중에서 선택된 아미노산 서열 또는 서열 73, 97, 및 119와 비교하여 1개, 2개, 3개, 4개 또는 5개의 아미노산 치환, 결실 또는 부가를 갖는 아미노산 서열을 갖는 VL CDR2 영역; 및 서열 74, 98, 및 120으로 이루어진 군 중에서 선택된 아미노산 서열 또는 서열 74, 98, 및 120과 비교하여 1개, 2개, 3개, 4개 또는 5개의 아미노산 치환, 결실 또는 부가를 갖는 아미노산 서열을 갖는 VL CDR3 영역으로 이루어진 단리된 프로펠러 3 모노클로날 항체, 또는 그의 단편을 제공한다.
대안적
프레임워크
또는
스캐폴드로의
항체 단편의
그라프팅
생성되는 폴리펩티드가 LRP6에 특이적으로 결합하는 1개 이상의 결합 영역을 포함하는 한, 매우 다양한 항체/이뮤노글로불린 프레임워크 또는 스캐폴드를 사용할 수 있다. 이러한 프레임워크 또는 스캐폴드는 인간 이뮤노글로불린의 5가지 주요 이디오타입 (idiotype) 또는 그의 단편을 포함하고, 바람직하게는 인간화 측면을 갖는 다른 동물 종의 이뮤노글로불린을 포함한다. 신규한 프레임워크, 스캐폴드 및 단편이 당업자에 의해 계속 발견되고 개발되고 있다.
한 측면에서, 본 발명은 본 발명의 CDR이 그라프팅될 수 있는 비-이뮤노글로불린 스캐폴드를 사용하여 비-이뮤노글로불린 기반 항체를 생성하는 것에 관한 것이다. 표적 LRP6 단백질 (예를 들어, 인간 및/또는 시노몰구스 LRP6)에 특이적인 결합 영역을 포함한다면 공지된 또는 미래의 비-이뮤노글로불린 프레임워크 및 스캐폴드가 사용될 수 있다. 공지된 비-이뮤노글로불린 프레임워크 또는 스캐폴드는 피브로넥틴 (컴파운드 쎄라퓨틱스, 인크. (Compound Therapeutics, Inc.), 미국 매사추세츠주 왈담), 안키린 (몰레큘라 파트너즈 아게 (Molecular Partners AG), 스위스 취리히), 도메인 항체 (도만티스, 리미티드 (Domantis, Ltd.), 미국 매사추세츠주 캠브릿지, 및 아블링스 엔브이 (Ablynx nv), 벨기에 즈뷔나아르드), 리포칼린 (피에리스 프로테오랩 아게 (Pieris Proteolab AG), 독일 프라이징), 작은 모듈형 면역-의약품 (트루비온 파마슈티칼즈 인크. (Trubion Pharmaceuticals Inc.), 미국 워싱턴주 시애틀), 맥시바디 (아비디아, 인크. (Avidia, Inc.), 미국 캘리포니아주 마운틴뷰), 단백질 A (애피바디 아게 (Affibody AG), 스웨덴) 및 애필린 (감마-크리스탈린 또는 유비퀴틴) (스실 프로테인즈 게엠베하 (Scil Proteins GmbH), 독일 할레)을 포함하고 이로 제한되지 않는다.
피브로넥틴 스캐폴드는 피브로넥틴 유형 III 도메인 (예를 들어, 피브로넥틴 유형 III의 10번째 모듈 (10Fn3 도메인))을 기초로 한다. 피브로넥틴 유형 III 도메인은 단백질의 코어를 형성하도록 서로에 대해 패킹된 2개의 베타 시트 사이에 분포된 7개 또는 8개의 베타 가닥을 갖고, 베타 가닥들을 서로 연결하고 용매 노출된 루프 (CDR과 유사)를 추가로 함유한다. 베타 시트 샌드위치의 각 가장자리에는 이러한 루프가 적어도 3개 존재하고, 여기서 상기 가장자리는 베타 가닥의 방향에 수직인 단백질의 경계이다 (미국 6,818,418 참조). 이러한 피브로넥틴-기반 스캐폴드는 이뮤노글로불린은 아니지만, 전반적인 폴드 (fold)는 낙타 및 라마 IgG 내의 전체 항원 인식 단위를 포함하는 중쇄 가변 영역인 최소의 기능적 항체 단편의 것과 밀접한 관련이 있다. 이러한 구조로 인해, 비-이뮤노글로불린 항체는 항체의 것과 성질 및 친화도에서 유사한 항원 결합 특성을 모방한다. 이러한 스캐폴드는 생체내 항체의 친화도 성숙 과정과 유사한 시험관내 루프 무작위화 및 셔플링 (shuffling) 전략에서 사용될 수 있다. 이러한 피브로넥틴-기반 분자는 표준 클로닝 기술을 이용하여 분자의 루프 영역이 본 발명의 CDR로 대체될 수 있는 스캐폴드로 사용될 수 있다.
안키린 기술은 상이한 표적들에 대한 결합에 사용될 수 있는 가변 영역을 보유하도록 안키린 유래의 반복 모듈을 갖는 단백질을 스캐폴드로서 사용하는 것을 기초로 한다. 안키린 반복 모듈은 2개의 역평행한 α-나선 및 β-턴 (turn)으로 이루어진 33개 아미노산의 폴리펩티드이다. 가변 영역의 결합은 리보좀 디스플레이를 사용함으로써 대부분 최적화된다.
아비머는 천연 A-도메인 함유 단백질, 예컨대 LRP6에서 유래된다. 이러한 도메인은 본래 단백질-단백질 상호작용을 위해 사용되고, 인간에서는 250종 초과의 단백질이 구조적으로 A-도메인을 기초로 한다. 아비머는 아미노산 링커를 통해 연결된 많은 상이한 "A-도메인" 단량체들 (2개 내지 10개)로 이루어진다. 예를 들어 미국 특허 출원 공개 번호 20040175756; 20050053973; 20050048512; 및 20060008844에 기재된 방법을 이용하여 표적 항원에 결합할 수 있는 아비머가 생성될 수 있다.
애피바디 친화도 리간드는 단백질 A의 IgG-결합 도메인 중 하나의 스캐폴드를 기초로 하는 3개의 나선 다발로 이루어진 작고 단순한 단백질이다. 단백질 A는 박테리아 스타필로코쿠스 아우레우스 (Staphylococcus aureus)로부터의 표면 단백질이다. 이 스캐폴드 도메인은 58개의 아미노산으로 이루어지며, 이중 13개는 무작위화되어 다수의 리간드 변이체를 갖는 애피바디 라이브러리를 생성한다 (예를 들어, 미국 5,831,012 참조). 애피바디 분자는 항체를 모방하고, 항체의 분자량이 150 kDa인데 비해 분자량은 6 kDa이다. 애피바디 분자는 작은 크기에도 불구하고 이것의 결합 부위는 항체의 결합 부위와 유사하다.
안티칼린은 피에리스 프로테오랩 아게라는 회사가 개발한 제품이다. 이것은 화학적으로 감수성이 있거나 불용성인 화합물의 생리적인 수송 또는 저장에 일반적으로 관련되는 광범위한 군의 작고 강한 단백질인 리포칼린으로부터 유래된다. 여러 가지 천연 리포칼린이 인간 조직 또는 체액에서 생성된다. 단백질 구조는 초가변 루프가 단단한 프레임워크의 최상부에 존재하는 이뮤노글로불린을 연상시킨다. 그러나, 항체 또는 그의 재조합 단편과는 대조적으로, 리포칼린은 160개 내지 180개 아미노산 잔기의 단일 폴리펩티드 사슬로 이루어져, 단일 이뮤노글로불린 도메인보다 단지 약간만 더 크다. 결합 포켓을 이루는, 4개 루프의 세트는 두드러진 구조적 유연성을 나타내고 다양한 측쇄를 허용한다. 따라서, 결합 부위는 독점적인 방법에서는 상이한 형태의 규정된 표적 분자들을 높은 친화도 및 특이성으로 인식하도록 재성형될 수 있다. 리포칼린 패밀리의 한 단백질인, 피에리스 브라시카에 (Pieris Brassicae)의 빌린-결합 단백질 (BBP)이 4개 루프 세트의 돌연변이 유발에 의해 안티칼린을 개발하는데 사용되어 왔다. 안티칼린을 설명하고 있는 특허 출원의 한 예는 PCT 공개 번호 WO 199916873이다.
애필린 분자는 단백질 및 소분자에 대한 특이적 친화도를 위해 설계된 작은 비-이뮤노글로불린 단백질이다. 신규 애필린 분자는 상이한 인간 유래의 스캐폴드 단백질을 각각 기초로 하는 2개의 라이브러리로부터 매우 신속하게 선택될 수 있다. 애필린 분자는 이뮤노글로불린 단백질과 어떠한 구조적 상동성도 나타내지 않는다. 현재, 2종의 애필린 스캐폴드가 사용되는데, 이중 하나는 감마 결정질의 인간 구조적 눈 수정체 단백질이고, 다른 하나는 "유비퀴틴" 수퍼패밀리 단백질이다. 2종의 인간 스캐폴드는 모두 매우 작고, 고온 안정성을 나타내며, pH 변화 및 변성제에 거의 내성을 갖는다. 이러한 높은 안정성은 주로 단백질의 확장된 베타 시트 구조로 인한 것이다. 감마 결정질 유래 단백질의 예는 WO200104144에 기재되어 있고, "유비퀴틴-유사" 단백질의 예는 WO2004106368에 기재되어 있다.
단백질 에피토프 모방체 (PEM)는 단백질-단백질 상호작용에 관여하는 주요 2차 구조인 단백질의 베타-헤어핀 2차 구조를 모방하는 중간 크기의 시클릭 펩티드-유사 분자 (MW 1 내지 2 kDa)이다.
일부 실시양태에서, Fab는 Fc 영역을 변화시켜서 침묵 IgG1 포맷으로 전환된다. 예를 들어, 표 1의 항체 MOR08168, MOR08545, MOR06706, MOR06475, MOR08193, 및 MOR08473은 하기의 아미노산 서열을 첨가하고, 경쇄를 CS (경쇄가 람다인 경우) 또는 C (경쇄가 카파인 경우)로 치환함으로써 IgG1 포맷으로 전환될 수 있다.
본원에서 사용된 바와 같이, "침묵 IgG1"은 Fc-매개 이펙터 기능 (예를 들어, ADCC 및/또는 CDC)이 감소되도록 아미노산 서열이 변경된 IgG1 Fc 서열이다. 이러한 항체는 일반적으로 Fc 수용체에 대한 결합이 감소될 것이다. 몇몇의 다른 실시양태에서, Fab는 IgG2 포맷으로 전환된다. 예를 들어, 표 1의 항체 MOR08168, MOR08545, MOR06706, MOR06475, MOR08193, 및 MOR08473은 불변 서열을 하기의 IgG2의 중쇄에 대한 불변 서열로 치환함으로써 IgG2 포맷으로 전환될 수 있다.
인간 또는 인간화 항체
본 발명은 LRP6 단백질 (예를 들어, 인간 및/또는 시노몰구스 LRP6)에 특이적으로 결합하는 완전 인간 항체를 제공한다. 키메라 또는 인간화 항체와 비교할 때, 본 발명의 인간 LRP6 항체는 인간 대상체에게 투여될 때 더욱 감소된 항원성을 갖는다.
인간 LRP6 항체는 당업계에 공지되어 있는 방법을 이용하여 생성될 수 있다. 예를 들어, 비-인간 항체를 조작된 인간 항체로 전환시키는데 이용되는 인간공학 (humaneering) 기술이 있다. 미국 특허 공개 번호 20050008625는 비-인간 항체와 비교할 때 동일한 결합 특징을 유지하거나 또는 보다 양호한 결합 특징을 제공하면서 항체에서 비인간 항체 가변 영역을 인간 가변 영역으로 대체하는 생체내 방법을 기재하고 있다. 상기 방법은 비-인간 참조 항체의 가변 영역을 에피토프에 따라 완전 인간 항체로 대체하는 것을 기초로 한다. 생성되는 인간 항체는 일반적으로 참조 비인간 항체와 구조적으로 관련이 있지는 않지만, 참조 항체와 동일한 항원 상의 동일한 에피토프에 결합한다. 간략하게 설명하면, 일련의 에피토프-유도된 상보성 대체 방법은 항원에 대한 시험 항체의 결합에 반응하는 리포터 시스템의 존재 하에 제한된 양의 항원과의 결합에 대한 "경쟁자"와 참조 항체의 다양한 하이브리드의 라이브러리 ("시험 항체") 사이의 세포내 경쟁을 확립함으로써 가능하게 된다. 경쟁자는 참조 항체 또는 그의 유도체, 예컨대 단일 쇄 Fv 단편일 수 있다. 경쟁자는 또한 참조 항체와 동일한 에피토프에 결합하는, 항원의 천연 또는 인공 리간드일 수 있다. 경쟁자의 유일한 요건은 참조 항체와 동일한 에피토프에 결합하고, 항원 결합을 위해 참조 항체와 경쟁하는 것이다. 시험 항체는 비-인간 참조 항체로부터의 공통적인 1개의 항원 결합 V-영역, 및 다양한 공급원, 예컨대 인간 항체의 레퍼토리 라이브러리로부터 무작위로 선택된 다른 V-영역을 갖는다. 참조 항체로부터의 공통적인 V-영역은 가이드로서 기능하여, 시험 항체를 항원 상의 동일한 에피토프에 동일한 배향으로 위치시킴으로써, 선택이 참조 항체에 대해 가장 높은 항원 결합 충실도로 편향되도록 한다.
많은 유형의 리포터 시스템이 시험 항체와 항원 사이의 원하는 상호작용을 검출하는데 사용될 수 있다. 예를 들어, 시험 항체가 항원에 결합할 때에만 단편 상보성에 의한 리포터 활성화가 일어나도록 상보성 리포터 단편을 항원 및 시험 항체 각각에 연결할 수 있다. 시험 항체- 및 항원-리포터 단편 융합체가 경쟁자와 동시 발현되는 경우, 리포터 활성화는 항원에 대한 시험 항체의 친화도에 비례하는, 경쟁자와 경쟁하는 시험 항체의 능력에 의존하게 된다. 사용될 수 있는 다른 리포터 시스템은 미국 특허 출원 일련 번호 10/208,730 (공개 번호 20030198971)에 개시된 바와 같은 자가억제된 리포터 재활성화 시스템 (RAIR)의 재활성화제 또는 미국 특허 출원 일련 번호 10/076,845 (공개 번호 20030157579)에 개시된 경쟁적 활성화 시스템을 포함한다.
일련의 에피토프 유도된 상보성 대체 시스템을 사용하여, 경쟁자, 항원 및 리포터 성분과 함께 단일 시험 항체를 발현하는 세포를 확인하는 선택을 수행한다. 이러한 세포에서, 각각의 시험 항체는 제한된 양의 항원과의 결합을 위해 경쟁자와 1:1로 경쟁한다. 리포터의 활성은 시험 항체에 결합된 항원의 양에 비례하고, 이는 다시 항원에 대한 시험 항체의 친화도 및 시험 항체의 안정성에 비례한다. 시험 항체는 처음에는 시험 항체로서 발현될 때 참조 항체의 활성에 대한 그의 활성을 기초로 하여 선택된다. 제1 라운드의 선택 결과는 "하이브리드" 항체의 세트이고, 그 각각은 참조 항체로부터의 동일한 비-인간 V-영역 및 라이브러리로부터의 인간 V-영역으로 이루어지고, 그 각각은 항원 상에서 참조 항체와 동일한 에피토프에 결합한다. 제1 라운드에서 선택된 하이브리드 항체 중 하나 이상은 항원에 대한 친화도가 참조 항체와 유사하거나 그보다 더 높을 것이다.
제2의 V-영역 대체 단계에서, 제1 단계에서 선택된 인간 V-영역이 나머지 비-인간 참조 항체 V-영역을 동족 인간 V-영역의 다양한 라이브러리로 인간 대체하기 위한 선택의 가이드로서 사용된다. 제1 라운드에서 선택된 하이브리드 항체는 또한 제2 라운드의 선택을 위해 경쟁자로서 사용될 수도 있다. 제2 라운드의 선택 결과는, 참조 항체와 구조적으로 상이하지만 동일 항원과의 결합을 위해 참조 항체와 경쟁하는 완전 인간 항체의 세트이다. 선택된 인간 항체 중 일부는 참조 항체와 동일한 항원 상의 동일한 에피토프에 결합한다. 이들 선택된 인간 항체 중에서, 1종 이상은 참조 항체와 동등하거나 그보다 더 높은 친화도로 동일한 에피토프에 결합한다.
상기 기재된 마우스 또는 키메라 LRP6 항체 중의 하나를 참조 항체로서 사용함으로써, 상기 방법은 동일한 결합 특이성 및 동일하거나 더 양호한 결합 친화도로 인간 LRP6에 결합하는 인간 항체를 생성하는데 쉽게 이용될 수 있다. 또한, 이러한 인간 LRP6 항체는 또한 인간 항체를 통상적으로 생산하는 회사, 예를 들어 칼로바이오스, 인크. (KaloBios, Inc.) (미국 캘리포니아주 마운틴 뷰)로부터 구입할 수 있다.
낙타류 (
camelid
) 항체
신세계 멤버, 예컨대 라마 종 (라마 파코스 (Lama paccos), 라마 글라마 (Lama glama) 및 라마 비쿠그나 (Lama vicugna))을 비롯하여 낙타 및 단봉 낙타 (카멜루스 박트리아누스 (Camelus bactrianus) 및 칼레루스 드로마데리우스 (Calelus dromaderius)) 패밀리의 멤버로부터 얻은 항체 단백질을 인간 대상체에 대해 크기, 구조적 복합도 및 항원성에 관한 특징을 규명하였다. 자연계에서 발견되는 바와 같이 이러한 포유동물 패밀리로부터의 특정 IgG 항체에는 경쇄가 없고, 따라서, 다른 동물로부터의 항체에서의 2개의 중쇄 및 2개의 경쇄를 갖는 일반적인 4쇄 4차 구조와 구조적으로 상이하다. PCT/EP93/02214 (1994년 3월 3일 공개된 WO 94/04678)를 참조한다.
VHH로서 확인된 작은 단일 가변 도메인인 낙타류 항체 영역은 "낙타류 나노바디"로 공지된 저분자량 항체-유래 단백질을 생성하는, 표적에 대해 고친화도를 갖는 작은 단백질을 얻도록 하는 유전자 조작으로 수득할 수 있다. 1998년 6월 2일 허여된 미국 특허 번호 5,759,808을 참조하고, 또한 문헌 [Stijlemans et al., (2004) J Biol Chem 279:1256-1261], [Dumoulin et al., (2003) Nature 424:783-788], [Pleschberger. et al., (2003) Bioconjugate Chem 14:440-448], [Cortez-Retamozo et al., (2002) Int J Cancer 89:456-62] 및 [Lauwereys et al., (1998) EMBO J 17:3512-3520]을 참조한다. 낙타류 항체 및 항체 단편의 조작된 라이브러리는 시판되고 있으며, 예를 들어 아블링스 (벨기에 겐트)가 시판한다. 비-인간 기원의 다른 항체와 같이, 낙타류 항체의 아미노산 서열을 재조합 방식으로 변경시켜 인간 서열과 보다 근접하게 유사한 서열을 수득할 수 있고, 즉, 나노바디를 "인간화"할 수 있다. 따라서, 인간에 대한 낙타류 항체의 천연적으로 낮은 항원성을 추가로 저하시킬 수 있다.
낙타류 나노바디는 분자량이 인간 IgG 분자의 대략 1/10이고, 상기 단백질은 단지 수 나노미터의 물리적 직경을 갖는다. 작은 크기가 갖는 결과 중 하나는 보다 큰 항체 단백질의 경우에는 기능적으로 보이지 않는 항원 부위에 결합하는 낙타류 나노바디의 능력이고, 즉, 낙타류 나노바디는 고전적인 면역학적 기술을 이용하여 달리 알 수 없는 항원을 검출하는 시약으로서 및 가능한 치료제로서 유용하다. 따라서, 작은 크기가 갖는 또 다른 결과는, 낙타류 나노바디는 표적 단백질의 홈 또는 좁은 틈새 내의 특이적 부위에 대한 결합의 결과로 억제할 수 있기 때문에 고전적인 항체의 기능보다 고전적인 저분자량 약물의 기능과 보다 밀접하게 유사한 능력으로 작용할 수 있다는 것이다.
이러한 저분자량 및 조밀한 크기 때문에, 극도로 열 안정적이고 극한 pH 및 단백질 분해 소화에 대해서도 안정하고 낮은 항원성을 갖는 낙타류 나노바디가 추가로 생성된다. 또 다른 결과는 낙타류 나노바디가 순환계로부터 조직 내로 쉽게 이동하고 심지어는 혈액-뇌 장벽을 횡단하며, 신경 조직에 영향을 미치는 장애를 치료할 수 있다는 것이다. 나노바디는 또한 혈액 뇌 장벽을 횡단하는 약물 수송을 추가로 용이하게 할 수 있다. 2004년 8월 19일 공개된 미국 특허 출원 20040161738을 참조한다. 인간에 대한 낮은 항원성과 조합된 이러한 특징은 높은 치료 가능성을 나타낸다. 추가로, 이들 분자는 원핵 세포, 예를 들어 이. 콜라이 내에서 완전하게 발현될 수 있고, 박테리오파지와의 융합 단백질로서 발현되며, 기능적이다.
따라서, 본 발명의 특징은 LRP6에 대해 높은 친화도를 갖는 낙타류 항체 또는 나노바디이다. 본원의 특정 실시양태에서, 낙타류 항체 또는 나노바디는 낙타류 동물에서 천연적으로 생성되고, 즉, 다른 항체에 대해 본원에 기재된 기술을 이용하여 LRP6 또는 그의 펩티드 단편으로 면역화시킨 후 낙타류에 의해 생성된다. 별법으로, LRP6 낙타류 나노바디가 조작되고, 즉, 예를 들어 본원의 실시예에 기재된 바와 같이 표적으로서 LRP6를 사용한 패닝 절차를 이용하여 적절하게 돌연변이시킨 낙타류 나노바디 단백질을 디스플레이하는 파지의 라이브러리로부터 선택하여 생성된다. 조작된 나노바디는 수용 대상체 내에서의 반감기가 45분 내지 2주가 되도록 유전자 조작함으로써 추가로 맞춤 제조될 수 있다. 특정 실시양태에서, 낙타류 항체 또는 나노바디는 예를 들어 PCT/EP93/02214에 기재된 바와 같이 본 발명의 인간 항체의 중쇄 또는 경쇄의 CDR 서열을 나노바디 또는 단일 도메인 항체 프레임워크 서열로 그라프팅함으로써 수득된다.
다가 항체
본 발명은 하나 이상의 표적(들) 수용체 상의 2개의 상이한 결합 부위에 대한 적어도 2개의 수용체 결합 도메인을 포함하는 다가 항체 (예를 들어, 이중파라토프, 이중특이적 항체)를 특징으로 한다. 임상 이점은 하나의 항체 내의 2개 이상의 결합 특이성의 결합에 의해 제공될 수 있다 ([Morrison et al., (1997) Nature Biotech. 15:159-163]; [Alt et al. (1999) FEBS Letters 454:90-94]; [Zuo et al., (2000) Protein Engineering 13:361-367]; [Lu et al., (2004) JBC 279:2856-2865]; [Lu et al., (2005) JBC 280:19665-19672]; [Marvin et al., (2005) Acta Pharmacologica Sinica 26:649-658]; [Marvin et al., (2006) Curr Opin Drug Disc Develop 9:184-193]; [Shen et al., (2007) J Immun Methods 218:65-74]; [Wu et al., (2007) Nat Biotechnol. 11:1290-1297]; [Dimasi et al., (2009) J. Mol Biol. 393:672-692]; 및 [Michaelson et al., (2009) mAbs 1:128-141].
본 발명은 다가 항체 (예를 들어, 단일 LRP6 이중파라토프 또는 이중특이적 항체)가 프로펠러 1 (예를 들어 Wnt1) 및 프로펠러 3 (예를 들어 Wnt3) 리간드-매개 신호 전달을 모두 억제하는 능력을 갖는다는 발견을 기초로 한다. 또한, 예기치 않게, 다가 항체 (예를 들어, 단일 LRP6 이중파라토프 또는 이중특이적 항체)는 유의한 Wnt 신호의 강화를 보이지 않는다. 다가 항체는 구분되는 LRP6 β-프로펠러 영역에 결합한다. 프로펠러 1 항체는 β-프로펠러 1 도메인에 결합하고, 프로펠러 1-의존성 Wnt, 예컨대 Wnt1, Wnt2, Wnt6, Wnt7A, Wnt7B, Wnt9, Wnt10A, Wnt10B를 차단하고, Wnt1 신호 전달 경로를 억제한다. 프로펠러 3 항체는 β-프로펠러 3 도메인에 결합하고, 프로펠러 3-의존성 Wnt, 예컨대 Wnt3a 및 Wnt3을 차단하고, Wnt3 신호 전달 경로를 억제한다. LRP6 항체는 프로펠러 1 및 프로펠러 3 리간드를 2개의 별개의 클래스로 분화시키고, LRP6 표적 수용체의 구분되는 에피토프에 결합한다. LRP6 항체의 단편 (예를 들어, Fab)의 전장 IgG 항체로의 전환은 또 다른 단백질, 예컨대 Wnt1 또는 Wnt3 리간드의 존재 하에 Wnt 신호를 강화 (향상)하는 항체를 생성한다.
다가 항체는 전통적인 항체에 비해 이점, 예를 들어, 표적 레퍼토리의 확대, 새로운 결합 특이성 보유, 효능 증가, 및 신호 강화 부재를 제공한다. 단일 LRP6 다가 항체는 동일한 세포 상의 단일 LRP6 표적 수용체 상의 다중 프로펠러 영영에 결합하고, Wnt 신호 전달을 억제할 수 있다. 한 실시양태에서, 다가 항체는 프로펠러 1, 프로펠러 2, 프로펠러 3, 및 프로펠러 4로 이루어진 군 중에서 선택된 β-프로펠러 영역의 임의의 조합에 결합한다. 한 실시양태에서, 다가 항체는 LRP6의 프로펠러 1 및 프로펠러 3 도메인에 결합한다. 따라서, 단일 LRP6 다가 항체는 다수의 β-프로펠러 영역에 결합하고 각각의 도메인에 의해 매개되는 Wnt 신호 전달을 억제함으로써 작용 효능을 증가시킨다. 예를 들어, 단일 LRP6 다가 항체는 각각 프로펠러 1 및 프로펠러 3 도메인에 결합함으로써 프로펠러 1 및 프로펠러 3 매개 Wnt 신호 전달을 모두 억제한다. 작용 효능 증가는 LRP6 다가 항체의 결합력 증가 또는 우수한 결합에 의한 것일 수 있다.
한 실시양태에서, 다가 항체는 scFv를 IgG 항체에 연결함으로써 생산된다. scFv 제조에 사용되는 VH 및 VL 도메인은 동일한 또는 상이한 항체로부터 유래될 수 있다. scFv는 적어도 1, 2, 3, 4, 5, 또는 6개의 CDR을 포함한다.
IgG 항체의 Fc 영역 및 scFv 단편은 많은 상이한 배향으로 함께 연결될 수 있다. 한 실시양태에서, scFv는 Fc 영역의 C-말단에 연결된다. 다른 실시양태에서, scFv는 Fc 영역의 N-말단에 연결된다. 다른 실시양태에서, scFv는 Fc 영역의 N-말단 및 C-말단 둘 모두에 연결된다. 또 다른 실시양태에서, scFv는 항체의 경쇄에 연결될 수 있다. 본 발명의 다가 항체는 표적 수용체의 다중 결합 부위에 동시에 결합할 수 있다. 본 발명의 다가 항체의 수용체 결합 도메인은 적어도 1, 2, 3, 4, 5, 6, 7, 8 또는 그 초과의 결합 부위에 결합할 수 있다. 각각의 수용체 결합 도메인은 동일한 결합 부위에 대해 특이적일 수 있다. 본 발명의 다가 항체는 동일한 표적 수용체, 예를 들어, LRP6의 β-프로펠러 1 도메인 또는 β-프로펠러 3 도메인 상의 구분되는 에피토프에 특이적인 하나 이상의 수용체 결합 도메인을 포함한다. 별법으로, 본 발명의 다가 항체는 상이한 표적 수용체, 예를 들어, LRP6 및 LRP6이 아닌 수용체, 예컨대 Erb, cmet, IGFR1, 스무든드, 및 노치 수용체 상의 에피토프에 특이적인 하나 이상의 수용체 결합 도메인을 포함한다.
또한, 본 발명의 다가 항체 내의 각각의 수용체 결합 도메인은 전통적인 항체에 비해 항원에 대한 상이한 (즉, 더 높거나 더 낮은) 친화도를 가질 수 있다.
한 실시양태에서, 본 발명의 다가 항체는 LRP6 표적 수용체 상의 제1 에피토프에 대한 적어도 하나의 수용체 결합 도메인 및 동일한 LRP6 표적 수용체 상의 제2 에피토프에 대한 제2 수용체 결합 도메인을 포함하는 이중파라토프 항체이다.
본 발명의 다가 항체는 항체의 적어도 하나의 CDR, 제시된 항체의 적어도 2개의 CDR, 또는 제시된 항체의 적어도 3개의 CDR, 제시된 항체의 적어도 4개의 CDR, 제시된 항체의 적어도 5개의 CDR, 또는 제시된 항체의 적어도 6개의 CDR을 포함한다. 본 발명의 다가 항체는 항체의 적어도 하나의 VH 도메인, 제시된 항체의 적어도 하나의 VL 도메인, 또는 항체의 적어도 하나의 VH 도메인 및 하나의 VL 도메인을 포함한다. scFv 분자는 VH-링커-VL 배향 또는 VL-링커-VH 배향으로 제작될 수 있다.
본 발명의 scFv 분자 또는 이를 포함하는 융합 단백질의 안정성은 통상적인 대조군 scFv 분자 또는 전장 항체의 생물물리적 특성 (예를 들어, 열 안정성)을 참조로 하여 평가할 수 있다. 한 실시양태에서, 본 발명의 다가 항체는 대조군 결합 분자 (예를 들어 통상적인 scFv 분자)보다 약 0.1, 약 0.25, 약 0.5, 약 0.75, 약 1, 약 1.25, 약 1.5, 약 1.75, 약 2, 약 2.5, 약 3, 약 3.5, 약 4, 약 4.5, 약 5, 약 5.5, 약 6, 약 6.5, 약 7, 약 7.5, 약 8, 약 8.5, 약 9, 약 9.5, 약 10, 약 11, 약 12, 약 13, 약 14, 또는 약 15℃ 더 높은 열 안정성을 갖는다.
scFv 분자는 최적화된 길이 및/또는 아미노산 조성을 갖는 scFv 링커를 포함한다. 본 발명의 바람직한 scFv 링커는 통상적인 항체에 비해 본 발명의 다가 항체의 열 안정성을 적어도 약 2℃ 또는 3℃ 개선한다. 한 실시양태에서, 본 발명의 다가 항체는 통상적인 항체에 비해 1℃ 개선된 열 안정성을 갖는다. 또 다른 실시양태에서, 본 발명의 다가 항체는 통상적인 항체에 비해 2℃ 개선된 열 안정성을 갖는다. 또 다른 실시양태에서, 본 발명의 다가 항체는 통상적인 항체에 비해 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15℃ 개선된 열 안정성을 갖는다. 비교는 예를 들어, 본 발명의 scFv 분자 및 선행 기술의 방법을 이용하여 제조된 scFv 분자 사이에서 또는 scFv 분자 및 그로부터 scFv VH 및 VL이 유래한 항체의 Fab 단편 사이에서 이루어질 수 있다. 열 안정성은 당업계에 공지된 방법을 이용하여 측정할 수 있고, 예를 들어, 한 실시양태에서, Tm을 측정할 수 있다. Tm의 측정 방법 및 다른 단백질 안정성의 결정 방법을 아래에서 보다 상세히 설명한다.
한 실시양태에서, scFv 링커는 아미노산 서열 (Gly4Ser)3으로 이루어지거나 또는 (Gly4Ser)4 서열을 포함한다. 다른 예시적인 링커는 (Gly4Ser)5 및 (Gly4Ser)6 서열을 포함하거나 이로 이루어진다. 본 발명의 scFv 링커의 길이는 상이할 수 있다. 한 실시양태에서, 본 발명의 scFv 링커는 약 5 내지 약 50개 아미노산의 길이이다. 또 다른 실시양태에서, 본 발명의 scFv 링커는 약 10 내지 약 40개 아미노산의 길이이다. 또 다른 실시양태에서, 본 발명의 scFv 링커는 약 15 내지 약 30개 아미노산의 길이이다. 또 다른 실시양태에서, 본 발명의 scFv 링커는 약 15 내지 약 20개 아미노산의 길이이다. 링커 길이의 변화는 활성을 보유하거나 향상시키고, 활성 연구에서 매우 우수한 효능을 제시할 수 있다. scFv 링커는 당업계에 공지된 기술을 사용하여 폴리펩티드 서열 내로 도입될 수 있다. 예를 들어, PCR 돌연변이 유발이 사용될 수 있다. 변형은 DNA 서열 분석에 의해 확인할 수 있다. 플라스미드 DNA는 생산되는 폴리펩티드의 안정한 생산을 위해 숙주 세포를 형질전환하기 위해 사용될 수 있다.
한 실시양태에서, 본 발명의 scFv 분자는 VH 도메인과 VL 도메인 사이에 산재하는 (Gly4Ser)3 또는 (Gly4Ser)4의 아미노산 서열을 갖는 scFv 링커를 포함하고, 여기서, VH 및 VL 도메인은 디술피드 결합에 의해 연결된다.
본 발명의 scFv 분자는 VL 도메인 내의 아미노산을 VH 도메인 내의 아미노산과 연결하는 적어도 하나의 디술피드 결합을 추가로 포함할 수 있다. 시스테인 잔기는 디술피드 결합을 제공하기 위해 필요하다. 디술피드 결합은 예를 들어 VL의 FR4 및 VH의 FR2를 연결하거나 또는 VL의 FR2 및 VH의 FR4를 연결하기 위해 본 발명의 scFv 분자에 포함될 수 있다. 디술피드 결합을 위한 예시적인 위치 (카바트 넘버링)는 VH의 43, 44, 45, 46, 47, 103, 104, 105, 및 106 및 VL의 42, 43, 44, 45, 46, 98, 99, 100, 및 101을 포함한다. VH 및 VL 도메인을 코딩하는 유전자의 변형은 당업계에 공지된 기술, 예를 들어, 부위-지정 돌연변이 유발을 사용하여 달성될 수 있다.
scFv에서의 돌연변이는 scFv의 안정성을 변경시키고, scFv에 돌연변이가 없는 다가 항체에 비해 돌연변이된 scFv를 포함하는 다가 항체의 전체 안정성을 개선한다. scFv에 대한 돌연변이는 실시예에 제시되는 바와 같이 생성될 수 있다. 돌연변이된 scFv의 안정성은 실시예에서 설명되는 바와 같이 척도, 예컨대 Tm, 온도 변성 및 온도 응집을 사용하여 비돌연변이된 scFv와 비교될 수 있다. 돌연변이체 scFv의 결합 능력은 ELISA와 같은 검정을 사용하여 결정할 수 있다.
한 실시양태에서, 본 발명의 다가 항체는 돌연변이된 scFv가 개선된 안정성을 다가 항체에 부여하도록 scFv에 적어도 하나의 돌연변이를 포함한다. 또 다른 실시양태에서, 본 발명의 다가 항체는 돌연변이된 scFv가 개선된 안정성을 다가 항체에 부여하도록 scFv에 적어도 1, 2, 3, 4, 5, 6, 7, 8, 9, 10개의 돌연변이를 포함한다. 또 다른 실시양태에서, 본 발명의 다가 항체는 돌연변이된 scFv가 개선된 안정성을 다가 항체에 부여하도록 scFv에 돌연변이의 조합을 포함한다.
다가 항체, 예컨대 본 발명의 이중파라토프 항체가 본원에 개시된다. 상기 이중파라토프 항체는 LRP6의 의 하나 이상의 에피토프에 결합한다. ScFv는 링커, 예컨대 GlySer 링커를 사용하여 예를 들어 힌지 영역에서 Fc 영역에 연결된다. 한 실시양태에서, 본 발명은 Fc의 CH3 영역에 연결된 (GlyGlySer)2 링커를 사용하여 LRP6의 β-프로펠러 3 도메인에 결합하는 scFv에 연결된, LRP6의 β-프로펠러 1 도메인에 결합하는 항체 또는 항원 결합 단편에 관한 것이다. 한 실시양태에서, LPR6 β-프로펠러 1 도메인에 결합하는 전장 IgG 항체가, LRP6 β-프로펠러 3 도메인에 결합하는 항체의 scFv 단편을 부착시키기 위해 사용된다.
다가 항체, 예컨대 본 발명의 이중파라토프 항체는 표 2에 제시된 중쇄 및 경쇄 서열의 임의의 조합을 사용하여 제작될 수 있다.
| 본 발명의 이중파라토프 구축물 | ||
| 구축물 | 중쇄 서열 번호 | 경쇄 서열 번호 |
| BiPa Fc에 부착된 프로펠러 1 IgG/프로펠러 3 scFv |
166, 171, 173, 175, 195, 201, 및 207 | 170, 193, 199 및 205 |
| 대안적인 BiPa 경쇄에 부착된 프로펠러 1 IgG/프로펠러 3 scFv |
177 | 181 |
| 역 BiPa Fc에 부착된 프로펠러 3 IgG/프로펠러 1 scFv |
187 및 189 | 185 |
따라서, 본 발명은 프로펠러 1 IgG 항체 및 프로펠러 3 scFv를 사용하여 제작된 이중파라토프 항체에 관한 것이다. 한 실시양태에서, 이중파라토프 항체는 서열 166, 171, 173, 175, 195, 201, 및 207로 이루어진 군 중에서 선택된 임의의 중쇄 서열; 및 서열 170, 193, 199, 및 205로 이루어진 군 중에서 선택된 임의의 경쇄 서열을 사용하여 제작된다. 한 실시양태에서, 이중파라토프 항체는 서열 166/170, 171/170, 173/170, 175/170, 201/199, 207/205, 및 195/193으로 이루어진 군 중에서 선택된 중쇄 및 경쇄 서열을 포함한다.
한 실시양태에서, 이중파라토프 항체는 중쇄 서열 166 및 경쇄 서열 170을 포함한다. 한 실시양태에서, 이중파라토프 항체는 중쇄 서열 171 및 경쇄 서열 170을 포함한다. 한 실시양태에서, 이중파라토프 항체는 중쇄 서열 173 및 경쇄 서열 170을 포함한다. 한 실시양태에서, 이중파라토프 항체는 중쇄 서열 175 및 경쇄 서열 170을 포함한다. 한 실시양태에서, 이중파라토프 항체는 중쇄 서열 201 및 경쇄 서열 199를 포함한다. 한 실시양태에서, 이중파라토프 항체는 중쇄 서열 207 및 경쇄 서열 205을 포함한다. 한 실시양태에서, 이중파라토프 항체는 중쇄 서열 195 및 경쇄 서열 193을 포함한다.
또 다른 실시양태에서, 이중파라토프 항체는 scFv가 IgG의 경쇄에 부착된 "대안적인 이중파라토프 항체"이다. 한 실시양태에서, 이중파라토프 항체는 중쇄 서열 177 및 경쇄 서열 181을 포함한다.
또 다른 실시양태에서, LPR6 β-프로펠러 3 도메인에 결합하는 전장 IgG 항체는 LRP6 β-프로펠러 1 도메인에 결합하는 항체의 scFv 단편을 부착시키기 위해 사용되고, "역 (reverse) 이중파라토프"로서 언급된다. 한 실시양태에서, 본 발명의 역 이중파라토프 항체는 프로펠러 3 IgG 항체 및 프로펠러 1 scFv를 사용하여 제작된다. 한 실시양태에서, 역 이중파라토프 항체는 서열 187 및 189로 이루어진 군 중에서 선택된 임의의 중쇄 서열; 및 서열 185의 경쇄 서열을 사용하여 제작된다. 한 실시양태에서, 역 이중파라토프 항체는 중쇄 서열 187 및 경쇄 서열 185를 포함한다. 한 실시양태에서, 역 이중파라토프 항체는 중쇄 서열 189 및 경쇄 서열 185를 포함한다.
또한, 본 발명은 69, 93, 및 115로 이루어진 군 중에서 선택된 아미노산 서열을 갖는 CDR1 서열, 70, 94, 및 116으로 이루어진 군 중에서 선택된 아미노산 서열을 갖는 CDR2 서열, 71, 95, 및 117로 이루어진 군 중에서 선택된 아미노산 서열을 갖는 CDR3 서열을 포함하는 중쇄 가변 영역; 72, 96, 및 118로 이루어진 군 중에서 선택된 아미노산 서열을 갖는 CDR1 서열, 73, 97, 및 119로 이루어진 군 중에서 선택된 아미노산 서열을 갖는 CDR2 서열, 74, 98 및 120으로 이루어진 군 중에서 선택된 아미노산 서열을 갖는 CDR3 서열을 포함하는 경쇄 경쇄 가변 영역과 조합된, 1, 21, 및 47로 이루어진 군 중에서 선택된 아미노산 서열을 갖는 CDR1 서열, 2, 22, 및 48로 이루어진 군 중에서 선택된 아미노산 서열을 갖는 CDR2 서열, 3, 23, 및 49로 이루어진 군 중에서 선택된 아미노산 서열을 갖는 CDR3 서열을 포함하는 중쇄 가변 영역; 4, 24, 및 50으로 이루어진 군 중에서 선택된 아미노산 서열을 갖는 CDR1 서열, 5, 25, 및, 51로 이루어진 군 중에서 선택된 아미노산 서열을 갖는 CDR2 서열, 6, 26, 및 52로 이루어진 군 중에서 선택된 아미노산 서열을 갖는 CDR3 서열을 포함하는 경쇄 경쇄 가변 영역을 갖는 이중파라토프 항체에 관한 것이고; 항체는 LRP6 (예를 들어, 인간 및/또는 시노몰구스 LRP6)에 결합하고, LRP6 생물학적 활성을 억제하고, 이것은 본원에서 설명되는 바와 같은 Wnt 리포터 유전자 검정 또는 Wnt 지정 신호 전달의 다른 척도 (예를 들어, LRP6 인산화, β-카테닌 안정화 및 핵 전위, 세포 증식/생존)에서 측정될 수 있다.
본 발명의 다가 항체에서 사용될 수 있는 항체는 인간, 뮤린, 키메라 및 인간화 모노클로날 항체이다.
본 발명의 다가 항체는 당업계에 공지된 방법을 사용하여 구성성분 수용체 결합 도메인들을 접합시킴으로써 제조할 수 있다. 예를 들어, 다가 항체의 각각의 수용체 결합 도메인은 별개로 생성한 후, 서로 접합될 수 있다. 수용체 결합 도메인이 단백질 또는 펩티드일 때, 다양한 커플링 또는 가교결합제가 공유 접합을 위해 사용될 수 있다. 가교결합제의 예는 단백질 A, 카르보디이미드, N-숙신이미딜-S-아세틸-티오아세테이트 (SATA), 5,5'-디티오비스(2-니트로벤조산) (DTNB), o-페닐렌디말레이미드 (oPDM), N-숙신이미딜-3-(2-피리딜디티오)프로피오네이트 (SPDP) 및 술포숙신이미딜 4-(N-말레이미도메틸) 시클로헥산-1-카르복실레이트 (술포-SMCC) (예를 들어, 문헌 [Karpovsky et al., (1984) J. Exp. Med. 160:1686], [Liu et al., (1985) Proc. Natl. Acad. Sci. USA 82:8648] 참조)를 포함한다. 다른 방법은 문헌 [Paulus (1985) Behring Ins. Mitt. No. 78:118-132], [Brennan et al., (1985) Science 229:81-83] 및 [Glennie et al., (1987) J. Immunol. 139: 2367-2375]에 기재된 것들을 포함한다. 접합제는 SATA 및 술포-SMCC (둘 다 피어스 케미컬 컴퍼니 (Pierce Chemical Co.) (미국 일리노이주 록포드)로부터 이용가능함)이다.
수용체 결합 도메인이 항체인 경우, 이것들은 2개의 중쇄의 C-말단 힌지 영역을 술프히드릴 결합에 의해 접합될 수 있다. 특정 실시양태에서, 힌지 영역은 접합 전에 홀수의 술프히드릴 잔기, 예를 들어 1개의 술프히드릴 잔기를 함유하도록 변형된다.
별법으로, 수용체 결합 도메인은 동일 벡터 내에서 코딩될 수 있고, 동일 숙주 세포 내에서 발현 및 조립될 수 있다. 이 방법은 다가 항체가 mAb x mAb, mAb x Fab, Fab x F(ab')2 또는 리간드 x Fab 융합 단백질인 경우에 특히 유용하다. 이중특이적 분자의 제조 방법은 예를 들어 미국 특허 번호 5,260,203; 미국 특허 번호 5,455,030; 미국 특허 번호 4,881,175; 미국 특허 번호 5,132,405; 미국 특허 번호 5,091,513; 미국 특허 번호 5,476,786; 미국 특허 번호 5,013,653; 미국 특허 번호 5,258,498; 및 미국 특허 번호 5,482,858에 기재되어 있다.
다가 항체가 그의 표적에 대한 결합은 예를 들어 효소-연결 면역흡착 검정 (ELISA), 방사성 면역검정 (REA), FACS 분석, 생물학적 검정 (예를 들어, 성장 억제) 또는 웨스턴 블롯 검정으로 확인할 수 있다. 이들 검정은 각각 일반적으로 특히 관심이 있는 단백질-항체 복합체에 특이적인 표지된 시약 (예컨대, 항체)을 이용함으로써 상기 복합체의 존재를 검출한다.
또 다른 측면에서, 본 발명은 LRP6에 결합하는 본 발명의 항체의 2개 이상의 상이한 수용체 결합 도메인을 포함하는 다가 항체를 제공한다. 수용체 결합 도메인은 단백질 융합 또는 공유 또는 비-공유 연결을 통해 함께 연결될 수 있다. 예를 들어, 본 발명의 항체를 본 발명의 항체의 불변 영역, 예를 들어 Fc 또는 힌지 영역에 결합하는 항체와 가교결합시켜 4가 항체를 수득할 수 있다.
다가 항체 배향
본 발명은 예를 들어 항체 가변 영역, 항체 단편 (예를 들어, Fab), scFv, 단일 쇄 디아바디, 또는 IgG 항체를 포함하는 다수의 수용체 결합 도메인 ("RBD")을 갖는 다가 항체에 관한 것이다. RBD의 예는 Fab 단편의 성분인 VL, VH, CL 및 CH1 도메인으로 이루어지는 일가 단편; F(ab)2 단편, 즉 힌지 영역에서 디술피드 다리에 의해 연결된 2개의 Fab 단편을 포함하는 이가 단편; VH 및 CH1 도메인으로 이루어지는 Fd 단편; 항체의 단일 아암의 VL 및 VH 도메인으로 이루어지는 Fv 단편; VH 도메인으로 이루어지는 dAb 단편 (Ward et al., (1989) Nature 341:544-546); 및 단리된 상보성 결정 영역 (CDR)이다. 또한, RBD는 단일 도메인 항체, 맥시바디, 유니바디, 미니바디, 디아바디, 트리아바디, 테트라바디, v-NAR 및 비스(bis)-scFv의 성분이다 (예를 들어, 문헌 [Hollinger and Hudson, (2005) Nature Biotechnology 23: 1126-1136] 참조).
본 발명의 다가 항체는, 생성되는 다가 항체가 기능적 활성 (예를 들어, Wnt 신호 전달의 억제)을 보유하는 한, 적어도 하나의 수용체 결합 도메인 (예를 들어, scFv, 단일 쇄 디아바디, 항체 가변 영역)을 사용하여 임의의 배향으로 생성된다. 생성되는 다가 항체가 기능적 활성 (예를 들어, Wnt 신호 전달의 억제)을 보유하는 한, 임의의 수의 수용체 결합 도메인이 Fc의 C-말단 및/또는 N-말단에 첨가할 수 있음을 이해하여야 한다. 한 실시양태에서, 1, 2, 3개, 또는 그 초과의 수용체 결합 도메인이 Fc 영역의 C-말단에 연결된다. 다른 실시양태에서, 1, 2, 3개, 또는 그 초과의 수용체 결합 도메인이 Fc 영역의 N-말단에 연결된다. 다른 실시양태에서, 1, 2, 3개, 또는 그 초과의 수용체 결합 도메인이 Fc 영역의 N-말단 및 C-말단 모두에 연결된다. 예를 들어, 본 발명의 다가 항체는 Fc 영역의 C-말단 및/또는 N-말단에 연결된 동일한 유형의 하나 초과의 수용체 결합 도메인, 예를 들어, scFv-scFv-Fc-IgG을 포함할 수 있다. 별법으로, 본 발명의 다가 항체는 Fc 영역의 C-말단 및/또는 N-말단에 연결된 상일한 유형의 하나 초과의 수용체 결합 도메인, 예를 들어, scFv-디아바디-Fc-IgG를 포함할 수 있다. 또 다른 실시양태에서, 1, 2, 3개 또는 그 초과의 수용체 결합 도메인 (예를 들어, scFv)은 IgG의 C-말단에 연결된다. 또 다른 실시양태에서, 1, 2, 3개 또는 그 초과의 수용체 결합 도메인 (예를 들어, scFv)은 IgG의 N-말단에 연결된다. 또 다른 실시양태에서, 1, 2, 3개 또는 그 초과의 수용체 결합 도메인 (예를 들어, scFv)은 IgG의 N-말단 및 C-말단에 연결된다.
다른 실시양태에서, 본 발명의 다가 항체는 C-말단에 연결된 상이한 유형의 하나 초과의 수용체 결합 도메인, 예를 들어, scFv-디아바디-Fc-IgG; 디아바디-scFv-Fc-IgG; scFv-scFv-디아바디-Fc-IgG; scFv-디아바디-scFv-Fc-IgG; 디아바디-scFv-scFv-Fc-IgG; 항체 가변 영역-scFv-디아바디-Fc-IgG 등을 사용하여 생성된다. 수용체 결합 도메인의 임의의 수의 순열을 갖는 다가 항체가 생성될 수 있다. 상기 다가 항체는 본원에서 설명되는 방법 및 검정을 사용하여 기능성에 대해 시험될 수 있다.
다른 실시양태에서, 본 발명의 다가 항체는 N-말단에 연결된 상이한 유형의 하나 초과의 수용체 결합 도메인, 예를 들어, IgG-Fc-scFv-디아바디; IgG-Fc-디아바디-scFv; IgG-Fc-scFv-scFv-디아바디; IgG-Fc-scFv-디아바디-scFv; IgG-Fc-디아바디-scFv-scFv; IgG-Fc-항체 가변 영역-scFv-디아바디 등을 사용하여 생성된다.
또 다른 실시양태에서, 본 발명의 다가 항체는 Fc 영역의 C-말단 및 N-말단에 연결된 단일 수용체 결합 도메인 (예를 들어, scFv, 단일 쇄 디아바디, 항체 가변 영역)을 사용하여 생성된다. 또 다른 실시양태에서, 다수의 수용체 결합 도메인은 Fc 영역의 N-말단에 연결된다. 예를 들어,적어도 1, 2, 3, 4, 5, 6, 7, 8개 또는 그 초과의 수용체 결합 도메인이 Fc 영역의 C-말단 및 N-말단에 연결된다. 예를 들어, 본 발명의 다가 항체는 Fc 영역의 C-말단 및 N-말단에 연결된 하나 이상의 scFv, 예를 들어, scFv-Fc-scFv-scFv; scFv-scFv-Fc-scFv-scFv 등을 포함할 수 있다. 다른 실시양태에서, 본 발명의 다가 항체는 N-말단에 연결된 상이한 유형의 하나 초과의 수용체 결합 도메인, 예를 들어, scFv-Fc-scFv-디아바디; scFv-Fc-디아바디-scFv; scFv-Fc-scFv-scFv-디아바디; scFv-Fc-scFv-디아바디-scFv; scFv-Fc-디아바디-scFv-scFv; scFv-Fc-항체 가변 영역-scFv-디아바디 등을 사용하여 생성된다. 수용체 결합 도메인의 임의의 수의 순열을 갖는 다가 항체가 생성될 수 있다. 상기 다가 항체는 본원에서 설명되는 방법 및 검정을 사용하여 기능성에 대해 시험될 수 있다.
링커 길이
scFv의 가변 영역이 폴딩하고 상호작용하는 방식에 링커 길이가 영향을 줄 수 있음이 알려져 있다. 실제로, 짧은 링커 (예를 들어, 5-10개 아미노산; 5-20개 아미노산)가 사용될 경우, 사슬내 폴딩 (미스폴딩)이 억제되고, 2개의 가변 영역을 함께 모아서 기능적 에피토프 결합 부위를 형성하기 위해 사슬간 폴딩이 필요하다. 링커 배향 및 크기의 예에 대해서는, 예를 들어, 본원에 참조로 포함시킨 문헌 [Hollinger et al. 1993 Proc Natl Acad. Sci. U.S.A. 90:6444-6448], 미국 특허 출원 공개 번호 2005/0100543, 2005/0175606, 2007/0014794, 및 PCT 공개 번호 WO2006/020258 및 WO2007/024715를 참조한다.
또한, 수용체 결합 도메인은 다양한 길이의 링커 영역에 의해 분리될 수 있음이 이해된다. 수용체 결합 도메인은 링커 서열에 의해 서로, C카파/람다, CH1, 힌지, CH2, CH3, 또는 전체 Fc 영역으로부터 분리될 수 있다. 상기 링커 서열은 무작위 분류의 아미노산, 또는 제한된 세트의 아미노산을 포함할 수 있다. 상기 링커 서열은 가요성이거나 경질일 수 있다.
본 발명의 다가 항체는 하나 이상의 그의 수용체 결합 도메인, C카파/람다 도메인, CH1 도메인, 힌지 영역, CH2 도메인, CH3 도메인, 또는 Fc 영역 사이의 적어도 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75개, 또는 그 초과의 아미노산 잔기의 링커 서열을 포함한다. 링커 서열은 임의의 천연 생성 아미노산으로 이루어질 수 있다. 몇몇 실시양태에서, 아미노산 글리신 및 세린이 링커 서열 내의 아미노산을 구성한다. 또 다른 실시양태에서, 링커 영역 배향은 글리신 반복체 (Gly4Ser)n의 세트를 포함하고, 여기서, n은 1 이상의 양의 정수이다.
한 실시양태에서, 링커는 (Gly4Ser)4 또는 (Gly4Ser)3을 포함하고 이로 제한되지 않는다. 또 다른 실시양태에서, 링커 Glu 및 Lys 잔기가 보다 우수한 용해도를 위해 Gly-Ser 링커 내에 산재될 수 있다. 또 다른 실시양태에서, 링커는 (Gly2Ser), (GlySer) 또는 (Gly3Ser)의 다수의 반복체를 포함한다. 또 다른 실시양태에서, 링커는 (Gly3Ser)+(Gly4Ser)+(GlySer)의 조합 및 병렬을 포함한다. 또 다른 실시양태에서, Ser은 Ala으로 교체될 수 있는데, 예를 들어, (Gly4Ala) 또는 (Gly3Ala)일 수 있다. 또 다른 실시양태에서, 링커는 모티프 (GluAlaAlaAlaLys)n를 포함하고, n은 1 이상의 양의 정수이다.
힌지
영역
본 발명의 다가 항체는 항체 힌지 영역의 전부 또는 적어도 일부를 포함할 수 있다. 힌지 영역 또는 그의 일부는 수용체 결합 도메인, CH1, C카파/람다, CH2, 또는 CH3에 직접 연결될 수 있다. 한 실시양태에서, 힌지 영역, 또는 그의 일부는 가변 길이 링커 영역을 통해 수용체 결합 도메인, CH1, C카파/람다, CH2, 또는 CH3에 연결될 수 있다.
본 발명의 다가 항체는 1, 2, 3, 4, 5, 6개, 또는 그 초과의 힌지 영역 또는 그의 일부를 포함할 수 있다. 힌지 영역 또는 그의 일부는 동일하거나 상이할 수 있다. 한 실시양태에서, 본 발명의 다가 항체는 인간 IgG1 분자로부터의 힌지 영역 또는 그의 일부를 포함한다. 추가의 실시양태에서, 힌지 영역 또는 그의 일부는 천연 생성 시스테인 잔기를 제거하거나, 비-천연 생성 시스테인 잔기를 도입하거나, 또는 천연 생성 잔기로 비-천연 생성 시스테인 잔기를 대체하도록 조작될 수 있다. 몇몇 실시양태에서, 본 발명의 다가 항체는 EPKSCDKTHTCPPCP (서열 211) 또는 EPKSC (서열 212)의 아미노산 서열을 포함하는 적어도 하나의 힌지 영역 또는 그의 일부를 함유한다. 몇몇 실시양태에서, 적어도 하나의 힌지 영역 또는 그의 일부는 적어도 하나의 천연 생성 시스테인 잔기를 또 다른 아미노산 잔기로 교체하도록 조작된다. 몇몇 실시양태에서, 적어도 하나의 천연 생성 시스테인 잔기는 세린으로 교체된다.
부위-특이적 접합에 유용한 비-천연 생성 시스테인 잔기는 본 발명의 다가 항체 내로 조작된다. 상기 방법, 조성물 및 방법은 2008년 1월 18일 출원된 미국 특허 가출원 61/022,073 ("Cysteine Engineered Antibodies for Site-Specific Conjugation") 및 2005년 9월 22일 출원된 미국 특허 출원 공개 번호 20070092940에 예시되어 있고, 이들은 각각 모든 목적을 위해 그 전부가 본원에 참고로 포함된다.
단백질 안정성의 평가 방법
다가 항체의 안정성을 평가하기 위해, 다중도메인 단백질의 최소로 안정한 도메인의 안정성을 본 발명의 방법 및 아래에서 설명되는 것을 사용하여 예측한다.
상기 방법은 최소로 안정한 도메인이 먼저 비폴딩되거나 또는 협력하여 비폴딩되는 다중도메인 단위 (즉, 단일 비폴딩 전이체를 보이는 다중도메인 단백질)의 전체 안정성 역치를 제한하는 다수의 열적 비폴딩 전이체의 결정을 가능하게 한다. 최소로 안정한 도메인은 많은 추가의 방식으로 확인될 수 있다. 돌연변이 유발은 어떤 도메인이 전체 안정성을 제한하는지 조사하기 위해 수행될 수 있다. 추가로, 다중도메인 단백질의 프로테아제 저항성은 DSC 또는 다른 분광 방법을 통해 최소로 안정한 도메인이 본질적으로 비폴딩되는 것으로 알려진 조건 하에 수행될 수 있다 ([Fontana, et al., (1997) Fold. Des., 2:R17-26]; [Dimasi et al. (2009) J. Mol. Biol. 393:672-692]). 일단 최소로 안정한 도메인이 확인된 후, 상기 도메인 (또는 그의 일부)을 코딩하는 서열은 본 발명의 방법에서 시험 서열로서 사용될 수 있다.
a) 열 안정성
본 발명의 조성물의 열 안정성은 당업계에 공지된 많은 비-제한적인 생물물리적 또는 생화학적 기술을 사용하여 분석될 수 있다. 특정 실시양태에서, 열 안정성을 분석적 분광법에 의해 평가한다.
예시적인 분석 분광 방법은 시차 주사 열량분석 (Differential Scanning Calorimetry; DSC)이다. DSC는 대부분의 단백질 또는 단백질 도메인의 비폴딩에 수반되는 열 흡수에 감수성인 열량계를 사용한다 (예를 들어, 문헌 [Sanchez-Ruiz, et al., Biochemistry, 27:1648-52, 1988] 참조). 단백질의 열 안정성을 결정하기 위해, 단백질 샘플을 열량계 내에 삽입하고, Fab 또는 scFv가 비폴딩될 때까지 온도를 올린다. 단백질이 비폴딩되는 온도는 전체적인 단백질 안정성을 표시한다.
또 다른 예시적인 분석 분광 방법은 원평광 이색성 (CD) 분광법이다. CD 분광법은 증가하는 온도의 함수로서 조성물의 광학 활성을 측정한다. 원평광 이색성 (CD) 분광법은 구조적 비대칭에 의해 발생하는 좌선 편광 대 우선 편광의 흡수 차이를 측정한다. 무질서한 또는 비폴딩된 구조는 정연한 또는 비폴딩된 구조와 매우 상이한 CD 스펙트럼을 생성한다. CD 스펙트럼은 증가하는 온도의 변성 효과에 대한 단백질의 감수성을 반영하고, 따라서 단백질의 열 안정성을 표시한다 ([van Mierlo and Steemsma, J. Biotechnol., 79(3):281-98, 2000] 참조).
열 안정성을 측정하기 위한 또 다른 예시적인 분석 분광 방법은 형광 방출 분광법이다 ([van Mierlo and Steemsma, 상기 문헌] 참조). 열 안정성을 측정하기 위한 또 다른 예시적인 분석 분광 방법은 핵 자기 공명 (NMR) 분광법이다 (예를 들어 [van Mierlo and Steemsma, 상기 문헌] 참조).
본 발명의 조성물의 열 안정성은 생화학적으로 측정할 수 있다. 열 안정성을 평가하기 위한 예시적인 생화학적 방법은 열 챌린지 (challenge) 검정이다. "열 챌린지 검정"에서, 본 발명의 조성물은 일정 시간 동안 다양한 승온에 적용된다. 예를 들어, 한 실시양태에서, 시험 scFv 분자, 또는 scFv 분자를 포함하는 분자는 예를 들어 1-1.5시간 동안 다양한 승온에 적용된다. 이어서, 단백질의 활성을 관련된 생화학적 검정에 의해 검정한다. 예를 들어, 단백질이 결합 단백질 (예를 들어 본 발명의 scFv 또는 scFv-함유 폴리펩티드)일 경우, 결합 단백질의 결합 활성은 기능적 또는 정량적 ELISA에 의해 결정될 수 있다.
상기 검정은 고처리량 (high throughput) 포맷 및 이. 콜라이 및 고처리량 스크리닝을 사용하는 실시예에 개시된 방식으로 수행할 수 있다. scFv 변이체의 라이브러리는 당업계에 공지된 방법을 사용하여 생성할 수 있다. scFv 발현은 유도될 수 있고, scFv는 열 챌린지에 적용될 수 있다. 챌린지 시험 샘플은 결합에 대해 검정될 수 있고, 안정한 scFv는 규모 확대되어 추가로 특성 결정될 수 있다.
열 안정성은 임의의 상기 기술 (예를 들어 분석 분광 기술)을 사용하여 본 발명의 조성물의 용융 온도 (Tm)를 측정함으로써 평가한다. 용융 온도는 조성물의 50%의 분자가 폴딩된 상태로 존재하는 열 전이 곡선의 중간지점의 온도이다 (예를 들어, 문헌 [Dimasi et al. (2009) J. Mol Biol. 393:672-692] 참조). 한 실시양태에서, scFv에 대한 Tm 값은 약 40℃, 41℃, 42℃, 43℃, 44℃, 45℃, 46℃, 47℃, 48℃, 49℃, 50℃, 51℃, 52℃, 53℃, 54℃, 55℃, 56℃, 57℃, 58℃, 59℃, 60℃, 61℃, 62℃, 63℃, 64℃, 65℃, 66℃, 67℃, 68℃, 69℃, 70℃, 71℃, 72℃, 73℃, 74℃, 75℃, 76℃, 77℃, 78℃, 79℃, 80℃, 81℃, 82℃, 83℃, 84℃, 85℃, 86℃, 87℃, 88℃, 89℃, 90℃, 91℃, 92℃, 93℃, 94℃, 95℃, 96℃, 97℃, 98℃, 99℃, 100℃이다. 한 실시양태에서, IgG에 대한 Tm 값은 약 40℃, 41℃, 42℃, 43℃, 44℃, 45℃, 46℃, 47℃, 48℃, 49℃, 50℃, 51℃, 52℃, 53℃, 54℃, 55℃, 56℃, 57℃, 58℃, 59℃, 60℃, 61℃, 62℃, 63℃, 64℃, 65℃, 66℃, 67℃, 68℃, 69℃, 70℃, 71℃, 72℃, 73℃, 74℃, 75℃, 76℃, 77℃, 78℃, 79℃, 80℃, 81℃, 82℃, 83℃, 84℃, 85℃, 86℃, 87℃, 88℃, 89℃, 90℃, 91℃, 92℃, 93℃, 94℃, 95℃, 96℃, 97℃, 98℃, 99℃, 100℃이다. 한 실시양태에서, 다가 항체에 대한 Tm 값은 약 40℃, 41℃, 42℃, 43℃, 44℃, 45℃, 46℃, 47℃, 48℃, 49℃, 50℃, 51℃, 52℃, 53℃, 54℃, 55℃, 56℃, 57℃, 58℃, 59℃, 60℃, 61℃, 62℃, 63℃, 64℃, 65℃, 66℃, 67℃, 68℃, 69℃, 70℃, 71℃, 72℃, 73℃, 74℃, 75℃, 76℃, 77℃, 78℃, 79℃, 80℃, 81℃, 82℃, 83℃, 84℃, 85℃, 86℃, 87℃, 88℃, 89℃, 90℃, 91℃, 92℃, 93℃, 94℃, 95℃, 96℃, 97℃, 98℃, 99℃, 100℃이다.
열 안정성은 또한 분석 열량분석 기술 (예를 들어 DSC)을 사용하여 본 발명의 조성물의 비열 또는 열용량 (Cp)을 측정함으로써 평가한다. 조성물의 비열은 1 mol의 물의 온도를 1℃ 올리는데 필요한 에너지 (예를 들어, kcal/mol)이다. 큰 Cp는 변성된 또는 불활성 단백질 조성물의 특징이다. 조성물의 열용량의 변화 (ΔCp)는 그의 열 전이 전후의 조성물의 비열을 결정함으로써 측정된다. 열 안정성은 또한 비폴딩의 깁스 (Gibbs) 자유 에너지 (ΔG), 비폴딩의 엔탈피 (ΔH), 또는 비폴딩의 엔트로피 (ΔS)를 비롯한 다른 열역학적 안정성 파라미터를 측정하거나 결정함으로써 평가할 수 있다.
상기 생화학적 검정의 하나 이상 (예를 들어 열 챌린지 검정)을 사용하여 조성물의 50%가 그의 활성 (예를 들어 결합 활성)을 보유하는 온도 (즉, TC 값)을 결정한다.
또한, scFv에 대한 돌연변이는 비돌연변이된 scFv에 비해 scFv의 열 안정성을 변경한다. 돌연변이된 scFv가 다가 항체 내로 도입되면, 돌연변이된 scFv는 전체 다가 항체에 열 안정성을 부여한다. 한 실시양태에서, scFv는 scFv에 열 안정성을 부여하는 단일 돌연변이를 포함한다. 또 다른 실시양태에서, scFv는 scFv에 열 안정성을 부여하는 다수의 돌연변이를 포함한다. 한 실시양태에서, scFv 내의 다수의 돌연변이는 열 scFv의 안정성에 대해 상가적 효과를 갖는다.
b) % 응집
본 발명의 조성물의 안정성은 그의 응집 경향을 측정함으로써 결정될 수 있다. 응집은 많은 비-제한적인 생화학적 또는 생물물리적 기술에 의해 측정될 수 있다. 예를 들어, 본 발명의 조성물의 응집은 크로마토그래피, 예를 들어 크기-배제 크로마토그래피 (SEC)를 이용하여 평가할 수 있다. SEC는 분자를 크기를 기초로 하여 분리한다. 칼럼을, 이온 및 소분자는 그 내부로 들어가도록 하고 큰 분자는 그렇지 않는 중합체 겔의 반고체 비드로 채운다. 단백질 조성물이 칼럼의 상부에 적용될 때, 치밀한 폴딩된 단백질 (즉, 비-응집된 단백질)은 큰 단백질 응집체에 이용가능한 것보다 더 큰 부피의 용매를 통해 분배된다. 그 결과, 큰 응집체는 칼럼을 통해 보다 신속하게 이동하고, 이러한 방식으로 혼합물은 그의 성분으로 분리되거나 분획화될 수 있다. 각각의 분획은 겔로부터 용리될 때 별개로 정량될 수 있다 (예를 들어 광 산란에 의해). 따라서, 본 발명의 조성물의 % 응집은 분획의 농도를 겔에 적용된 단백질의 총 농도와 비교함으로써 결정될 수 있다. 안정한 조성물은 본질적으로 단일 분획으로서 칼럼으로부터 용리되고, 용리 프로파일 또는 크로마토그램에서 본질적으로 단일 피크로서 나타난다.
c) 결합 친화도
본 발명의 조성물의 안정성은 그의 표적 결합 친화도를 결정함으로써 평가할 수 있다. 결합 친화도를 결정하기 위한 매우 다양한 방법이 당업계에 공지되어 있다. 결합 친화도를 결정하기 위한 예시적인 방법은 표면 플라스몬 공명을 이용한다. 표면 플라스몬 공명은 예를 들어 비아코어 시스템 (파마시아 바이오센서 에이비 (Pharmacia Biosensor AB), 스웨덴 웁살라 및 미국 뉴저지주 피스카타웨이)을 사용하여 바이오센서 매트릭스 내의 단백질 농도 변화의 검출에 의해 실시간 생물특이적 상호작용의 분석을 허용하는 광학 현상이다. 추가의 설명에 대해서는, 문헌 [Jonsson, U., et al. (1993) Ann. Biol. Clin. 51:19-26]; [Jonsson, U., i (1991) Biotechniques 11:620-627]; [Johnsson, B., et al. (1995) J. Mol. Recognit. 8:125-131]; 및 [Johnnson, B., et al. (1991) Anal. Biochem. 198:268-277]을 참조한다.
연장된 반감기를 갖는 항체
본 발명은 생체내 반감기가 연장된, LRP6 단백질에 특이적으로 결합하는 항체를 제공한다. 예를 들어, 신장 여과, 간에서의 대사, 단백질분해 효소 (프로테아제)에 의한 분화 및 면역원성 반응 (예를 들어, 항체에 의한 단백질 중화, 및 대식세포 및 수상돌기 세포에 의한 흡수)과 같은 많은 인자가 단백질의 생체내 반감기에 영향을 미칠 수 있다. 다양한 전략을 이용하여 본 발명의 항체의 반감기를 연장시킬 수 있다. 예를 들어, 폴리에틸렌글리콜 (PEG), reCODE PEG, 항체 스캐폴드, 폴리시알산 (PSA), 히드록시에틸 전분 (HES), 알부민-결합 리간드, 및 탄수화물 쉴드 (shield)로의 화학적 연결에 의해; 혈청 단백질, 예컨대 알부민, IgG, FcRn 및 트랜스페린에 결합하는 단백질과의 유전자 융합에 의해; 혈청 단백질, 예컨대 나노바디, Fab, DARPin, 아비머, 애피바디 및 안티칼린에 결합하는 다른 결합 모이어티와의 (유전적 또는 화학적) 커플링에 의해; rPEG, 알부민, 알부민의 도메인, 알부민-결합 단백질 및 Fc와의 유전자 융합에 의해; 또는 나노담체, 서방성 제형 또는 의료 장치 내로의 혼입에 의해 이루어진다.
항체의 생체내 혈청 순환을 연장시키기 위해서, 불활성 중합체 분자, 예컨대 고분자량 PEG를 다관능성 링커를 사용하거나 사용하지 않고 항체 또는 그의 단편에 항체의 N- 또는 C-말단에 대한 PEG의 부위 특이적 접합을 통해 또는 리신 잔기에 존재하는 엡실론-아미노기를 통해 부착시킬 수 있다. 항체를 PEG화 (pegylation)하기 위해, 항체 또는 그의 단편을 일반적으로 1개 이상의 폴리에틸렌 글리콜 (PEG) 기가 항체 또는 항체 단편에 부착되는 조건 하에 PEG, 예컨대 PEG의 반응성 에스테르 또는 알데히드 유도체와 반응시킨다. PEG화는 반응성 PEG 분자 (또는 유사한 반응성의 수용성 중합체)와의 아실화 반응 또는 알킬화 반응에 의해 수행될 수 있다. 본원에서 사용된 바와 같이, 용어 "폴리에틸렌 글리콜"은 다른 단백질을 유도체화하기 위해 사용된 임의의 형태의 PEG, 예컨대 모노 (C1-C10)알콕시- 또는 아릴옥시-폴리에틸렌 글리콜 또는 폴리에틸렌 글리콜-말레이미드를 포괄하는 것으로 의도된다. 특정 실시양태에서, PEG화되는 항체는 글리코실화되지 않은 항체이다. 생물학적 활성을 최소로 손실시키는 선형 또는 분지형 중합체 유도체화가 이용될 것이다. 항체에 대한 PEG 분자의 적당한 접합을 보장하기 위해서 접합 정도를 SDS-PAGE 및 질량 분광법으로 면밀히 모니터링할 수 있다. 미반응 PEG는 크기 배제 또는 이온 교환 크로마토그래피에 의해 항체-PEG 접합체로부터 분리될 수 있다. PEG-유도체화 항체는 당업자에게 공지된 방법, 예를 들어 본원에 기재된 면역검정을 이용하여 결합 활성 및 생체내 효능에 대해 시험될 수 있다. 단백질을 PEG화하는 방법은 당업계에 공지되어 있고, 본 발명의 항체에 적용할 수 있다. 예를 들어, EP 0 154 316 (니시무라(Nishimura) 등) 및 EP 0 401 384 (이시가와(Ishikawa) 등)를 참조한다.
다른 변형된 PEG화 기술은, 화학적으로 명시된 측쇄를 tRNA 합성효소 및 tRNA를 포함하는 재구축 시스템을 통해 생합성 단백질에 혼입하는, 화학적으로 직교 유도된 조작 (chemically orthogonal directed engineering) 기술의 재구축 (ReCODE PEG)을 포함한다. 상기 기술은 이. 콜라이, 효모 및 포유동물 세포에서 30개 초과의 새로운 아미노산을 생합성 단백질에 혼입할 수 있게 한다. tRNA는 앰버 (amber) 코돈이 위치한 임의의 부위에 비-천연 아미노산을 혼입시켜서, 정지 코돈의 앰버가 화학적으로 명시된 아미노산의 혼입 신호를 전달하는 코돈으로 전환되게 한다.
또한, 재조합 PEG화 기술 (rPEG)은 혈청 반감기 연장을 위해 사용될 수도 있다. 이 기술은 300개 내지 600개 아미노산의 비구조화 단백질 테일을 기존의 제약 단백질에 유전적으로 융합시키는 것을 포함한다. 이러한 비구조화 단백질 사슬의 겉보기 분자량은 그의 실제 분자량보다 약 15배 더 크기 때문에, 상기 단백질의 혈청 반감기는 크게 증가된다. 화학적 접합 및 재정제를 필요로 하는 통상적인 PEG화와는 달리, 제조 공정이 크게 단순화되고 생성물은 균일하다.
폴리시알화는 활성 수명을 연장하고 치료 펩티드 및 단백질의 안정성을 개선하기 위해서 천연 중합체 폴리시알산 (PSA)을 사용하는 또 다른 기술이다. PSA는 시알산의 중합체 (당)이다. 단백질 및 치료 펩티드 약물 전달을 위해 사용되는 경우, 폴리시알산은 접합체에 보호 미세환경을 제공한다. 이것은 순환계 내 치료 단백질의 활성 수명을 증가시키고, 이것이 면역계에 의해 인식되지 않도록 방지한다. PSA 중합체는 인간 신체에서 천연적으로 발견된다. 특정 박테리아는 수백만 년에 걸쳐서 자신의 벽을 이것으로 코팅하도록 진화하였다. 이후, 이러한 천연 폴리시알화 박테리아는 분자 모방에 의해 신체의 방어 시스템을 이겨낼 수 있었다. 자연계의 궁극적인 잠행 (stealth) 기술인 PSA는 이러한 박테리아로부터 대량으로 소정의 물리적 특징을 갖는 것으로서 쉽게 생산될 수 있다. 박테리아 PSA는 인체 내의 PSA와 화학적으로 동일하기 때문에 단백질에 커플링될 때조차도 완전하게 비-면역원성이다.
또 다른 기술은 항체에 연결된 히드록시에틸 전분 ("HES") 유도체의 사용을 포함한다. HES는 찰옥수수 전분에서 유래된 변형된 천연 중합체이며, 신체의 효소에 의해 대사될 수 있다. HES 용액은 일반적으로 부족한 혈액 부피를 채우고 혈액의 유동성을 개선하기 위해 투여된다. 항체의 HES화 (hesylation)는 상기 분자의 안정성을 증가시키고 또한 신장 청소율을 감소시켜서 순환 반감기의 연장을 가능하게 하여 생물학적 활성을 증가시킨다. 상이한 파리미터, 예컨대 HES의 분자량을 변경함으로써, 광범한 범위의 HES 항체 접합체가 맞춤 제조될 수 있다.
증가된 생체내 반감기를 갖는 항체는 또한 1개 이상의 아미노산 변형 (즉, 치환, 삽입 또는 결실)을 IgG 불변 도메인, 또는 그의 FcRn 결합 단편 (바람직하게는 Fc 또는 힌지 Fc 도메인 단편)에 도입하여 생성될 수 있다. 예를 들어, 국제 출원 공개 번호 WO 98/23289; 국제 출원 공개 번호 WO 97/34631; 및 미국 특허 번호 6,277,375를 참조한다.
추가로, 항체는 항체 또는 항체 단편을 생체 내에서 보다 안정적이고 보다 긴 생체내 반감기를 갖게 하도록 알부민에 접합시킬 수 있다. 상기 기술은 당업계에 공지되어 있으며, 예를 들어 국제 출원 공개 번호 WO 93/15199, WO 93/15200, 및 WO 01/77137; 및 유럽 특허 번호 EP 413,622를 참조한다.
LRP6 항체 또는 그의 단편은 또한 하나 이상의 인간 혈청 알부민 (HSA) 폴리펩티드, 또는 그의 일부에 융합될 수 있다. 그의 성숙 형태에서 585개 아미노산의 단백질인 HSA는 유의한 비율의 혈청 삼투압을 책임지고, 또한 내인성 및 외인성 리간드의 담체로서 기능한다. 담체 분자로서 알부민의 역할 및 그의 불활성 특성은 생체 내에서 폴리펩티드의 담체 및 수송체로서의 용도를 위해 바람직한 특성이다. 다양한 단백질의 담체로서의 알부민 융합 단백질의 성분으로서 알부민의 사용은 WO 93/15199, WO 93/15200, 및 EP 413 622에서 제안되었다. 또한, 폴리펩티드에 대한 융합을 위한 HSA의 N-말단 단편의 사용도 제안되었다 (EP 399 666). 따라서, 항체 또는 그의 단편을 유전적으로 또는 화학적으로 알부민에 융합 또는 접합시킴으로써 안정화 또는 저장 수명을 연장하고/하거나, 용액 내에서, 시험관 내에서 및/또는 생체 내에서 연장된 기간 동안 분자의 활성을 유지시킬 수 있다.
또 다른 단백질에 대한 알부민의 융합은 HSA를 코딩하는 DNA, 또는 그의 단편이 단백질을 코딩하는 DNA에 연결되도록 하는 유전자 조작에 의해 달성될 수 있다. 이어서, 융합 폴리펩티드를 발현하도록 적합한 플라스미드 상에 배열된 융합된 뉴클레오티드 서열로 적합한 숙주를 형질전환 또는 형질감염시킨다. 발현은 예를 들어 원핵 또는 진핵 세포로부터 시험관 내에서, 또는 생체 내에서, 예를 들어 트랜스제닉 유기체로부터 실시될 수 있다. HSA 융합에 관한 추가의 방법은 예를 들어 본원에 참조로 포함시킨 WO 2001077137 및 WO 200306007에서 찾을 수 있다. 특정 실시양태에서, 융합 단백질의 발현은 포유동물 세포주, 예를 들어, CHO 세포주에서 수행된다.
항체 접합체
본 발명은 이종 단백질 또는 폴리펩티드 (또는 그의 단편, 바람직하게는 적어도 10개, 적어도 20개, 적어도 30개, 적어도 40개, 적어도 50개, 적어도 60개, 적어도 70개, 적어도 80개, 적어도 90개 또는 적어도 100개 아미노산의 폴리펩티드)에 재조합 방식으로 융합되거나 화학적으로 접합 (공유 접합 및 비-공유 접합 둘 다를 포함함)되어 융합 단백질을 생성하는, LRP6 단백질에 특이적으로 결합하는 항체 또는 그의 단편을 제공한다. 특히, 본 발명은 본원에 기재된 항체 단편 (예를 들어, Fab 단편, Fd 단편, Fv 단편, F(ab)2 단편, VH 도메인, VH CDR, VL 도메인 또는 VL CDR) 및 이종 단백질, 폴리펩티드 또는 펩티드를 포함하는 융합 단백질을 제공한다. 단백질, 폴리펩티드 또는 펩티드를 항체 또는 항체 단편에 융합 또는 접합시키는 방법은 당업계에 공지되어 있다. 예를 들어, 미국 특허 번호 5,336,603, 5,622,929, 5,359,046, 5,349,053, 5,447,851, 및 5,112,946; 유럽 특허 번호 EP 307,434 및 EP 367,166; 국제 출원 공개 번호 WO 96/04388 및 WO 91/06570; [Ashkenazi et al., (1991) Proc. Natl. Acad. Sci. USA 88:10535-10539]; [Zheng et al., (1995) J. Immunol. 154:5590-5600]; 및 [Vil et al., (1992) Proc. Natl. Acad. Sci. USA 89:11337-11341]을 참조한다.
추가의 융합 단백질은 유전자-셔플링, 모티프-셔플링, 엑손-셔플링 및/또는 코돈-셔플링 (통칭하여, "DNA 셔플링"이라 지칭함)의 기술을 통해 생성될 수 있다. DNA 셔플링은 본 발명의 항체 또는 그의 단편 (예를 들어, 더 높은 친화도 및 더 낮은 해리 속도를 갖는 항체 또는 그의 단편)의 활성 변경에 사용될 수 있다. 일반적으로, 미국 특허 번호 5,605,793, 5,811,238, 5,830,721, 5,834,252, 및 5,837,458; 문헌 [Patten et al., (1997) Curr. Opinion Biotechnol. 8:724-33]; [Harayama, (1998) Trends Biotechnol. 16(2):76-82]; [Hansson et al., (1999) J. Mol. Biol. 287:265-76]; 및 [Lorenzo and Blasco, (1998) Biotechnology 24(2):308-313] (각각의 이들 특허 및 간행물은 그 전문이 본원에 참고로 포함됨)를 참조한다. 항체 또는 그의 단편, 또는 코딩된 항체 또는 그의 단편은 재조합 전에 오류-유발 (error-prone) PCR, 무작위 뉴클레오티드 삽입 또는 기타 방법에 의한 무작위 돌연변이 유발의 실시로 변경될 수 있다. LRP6 단백질에 특이적으로 결합하는 항체 또는 그의 단편을 코딩하는 폴리뉴클레오티드는 1종 이상의 이종 분자의 1종 이상의 성분, 모티프, 절편, 부분, 도메인, 단편 등과 재조합될 수 있다.
또한, 항체 또는 그의 단편은 마커 서열, 예컨대 정제를 용이하게 하는 펩티드에 융합될 수 있다. 바람직한 실시양태에서, 마커 아미노산 서열은 헥사-히스티딘 펩티드, 예컨대 특히 pQE 벡터 (퀴아젠, 인크. (QIAGEN, Inc., 미국 91311 캘리포니아주 채스워쓰 이튼 애비뉴 9259))에서 제공되는 태그이고, 많은 것들이 시판되고 있다. 예를 들어 문헌 [Gentz et al., (1989), Proc. Natl. Acad. Sci. USA 86:821-824]에 기재된 바와 같이, 헥사-히스티딘은 융합 단백질의 편리한 정제를 제공한다. 정제에 유용한 다른 펩티드 태그는 인플루엔자 혈구응집소 단백질로부터 유래된 에피토프에 대응하는 혈구응집소 ("HA") 태그 ([Wilson et al., (1984), Cell 37:767]), 및 "플래그 (flag)" 태그를 포함하고, 이로 제한되지 않는다.
다른 실시양태에서, 본 발명의 항체 또는 그의 단편은 진단제 또는 검출가능한 작용제에 접합된다. 이러한 항체는 특정 요법의 효능을 결정하는 것과 같은 임상 시험 절차의 일부로서 질환 또는 장애의 발병, 발생, 진행 및/또는 중증도를 모니터링하거나 예측하는데 유용할 수 있다. 이러한 진단 및 검출은 항체를 검출가능한 물질, 예를 들어 다양한 효소, 예컨대 (이에 제한되지 않음) 양고추냉이 퍼옥시다제, 알칼리성 포스파타제, 베타-갈락토시다제 또는 아세틸콜린에스테라제 (이에 제한되지 않음); 보결 분자단, 예컨대 스트렙타비딘/비오틴 및 아비딘/비오틴 (이에 제한되지 않음); 형광 물질, 예컨대 움벨리페론, 플루오레세인, 플루오레세인 이소티오시아네이트, 로다민, 디클로로트리아지닐아민 플루오레세인, 단실 클로라이드 또는 피코에리트린 (이에 제한되지 않음); 발광 물질, 예컨대 루미놀 (이에 제한되지 않음); 생체발광 물질, 예컨대 루시퍼라제, 루시페린 및 애쿠오린 (이에 제한되지 않음); 방사성 물질, 예컨대 아이오딘 (131I, 125I, 123I 및 121I), 탄소 (14C), 황 (35S), 삼중수소 (3H), 인듐 (115In, 113In, 112In 및 111In), 테크네튬 (99Tc), 탈륨 (201Ti), 갈륨 (68Ga, 67Ga), 팔라듐 (103Pd), 몰리브덴 (99Mo), 크세논 (133Xe), 불소 (18F), 153Sm, 177Lu, 159Gd, 149Pm, 140La, 175Yb, 166Ho, 90Y, 47Sc, 186Re, 188Re, 142Pr, 105Rh, 97Ru, 68Ge, 57Co, 65Zn, 85Sr, 32P, 153Gd, 169Yb, 51Cr, 54Mn, 75Se, 113Sn 및 117Tin (이에 제한되지 않음), 및 양전자 방출 금속 (다양한 양전자 방출 단층촬영술을 이용함), 및 비-방사성 상자성 금속 이온에 커플링시켜 달성될 수 있다.
본 발명은 추가로 치료 모이어티에 접합된 항체 또는 그의 단편의 용도를 포함한다. 항체 또는 그의 단편은 치료 모이어티, 예컨대 세포독소, 예를 들어, 세포증식 억제제 또는 세포파괴제, 치료제 또는 방사성 금속 이온, 예를 들어, 알파-방출체에 접합될 수 있다. 세포독소 또는 세포독성제는 세포에 유해한 임의의 작용제를 포함한다.
또한, 항체 또는 그의 단편은 주어진 생물학적 반응을 변형시키는 치료 모이어티 또는 약물 모이어티에 접합될 수 있다. 치료 모이어티 또는 약물 모이어티는 고전적인 화학 치료제로 제한되는 것으로 간주되지 않는다. 예를 들어, 약물 모이어티는 목적하는 생물학적 활성을 보유하는 단백질, 펩티드 또는 폴리펩티드일 수 있다. 이러한 단백질은 예를 들어 독소, 예컨대 아브린, 리신 A, 슈도모나스 (Pseudomonas) 외독소, 콜레라 독소 또는 디프테리아 독소; 단백질, 예컨대 종양 괴사 인자, α-인터페론, β-인터페론, 신경 성장 인자, 혈소판 유래 성장 인자, 조직 플라스미노겐 활성화제, 세포자멸제, 항-혈관신생제, 또는 생물학적 반응 변형제, 예를 들어 림포카인을 포함할 수 있다.
한 실시양태에서, 항-LRP6 항체, 또는 그의 단편은 치료적 모이어티, 예컨대 세포독소, 약물 (예를 들어, 면역억제제) 또는 방사성 독소에 접합된다. 상기 접합체는 본원에서 "면역접합체"로 언급된다. 하나 이상의 세포독소를 포함하는 면역접합체는 "면역독소"로서 언급된다. 세포독소 또는 세포독성제는 세포에 유해한 (예를 들어, 치사시키는) 임의의 작용제를 포함한다. 그 예는 탁손, 시토칼라신 B, 그라미시딘 D, 에티듐 브로마이드, 에메틴, 미토마이신, 에토포시드, 테노포시드, 빈크리스틴, 빈블라스틴, 티. 콜히친, 독소루비신, 다우노루비신, 히디드록시 안트라신 디온, 미톡산트론, 미트라마이신, 악티노마이신 D, 1-데히드로테스토스테론, 글루코코르티코이드, 프로카인, 테트라카인, 리도카인, 프로프라놀올, 및 퓨로마이신 및 그의 유사체 또는 상동체를 포함한다. 치료제는 또한 예를 들어 항대사물질 (예를 들어, 메토트렉세이트, 6-메르캅토퓨린, 6-티오구아닌, 시타라빈, 5-플루오로우라실 데카르바진), 제거제 (ablating agent) (예를 들어, 메클로르에타민, 티오테파 클로람부실, 멜팔란, 카르무스틴 (BSNU) 및 로무스틴 (CCNU), 시클로포스파미드, 부술판, 디브로모만니톨, 스트렙토조토신, 미토마이신 C, 및 시스-디클로로디아민 백금 (II) (DDP) 시스플라틴, 안트라사이클린 (예를 들어, 다우노루비신 (이전의 다우노마이신) 및 독소루비신), 항생제 (예를 들어, 닥티노마이신 (이전의 악티노마이신), 블레오마이신, 미트라마이신, 및 안트라마이신 (AMC)), 및 항-유사분열제 (예를 들어, 빈크리스틴 및 빈블라스틴)를 포함한다 (예를 들어, US20090304721 (시애틀 제네틱스(Seattle Genetics)) 참조).
본 발명의 항체에 접합될 수 있는 치료적 세포독소의 다른 예는 듀오카르마이신, 칼리케아미신, 메이탄신 및 오리스타틴, 및 이들의 유도체를 포함한다. 칼리케아미신 항체 접합체의 예는 상업상 이용가능하다 (밀로타르그 (Mylotarg)™; 와이어쓰-아이어스트 (Wyeth-Ayerst)).
세포독소는 당업계에서 이용가능한 링커 기술을 사용하여 본 발명의 항체에 접합될 수 있다. 세포독소를 항체에 접합시키기 위해 사용된 링커 종류의 예는 히드라존, 티오에테르, 에스테르, 디술피드 및 펩티드-함유 링커를 포함하고 이로 제한되지 않는다. 예를 들어 리소솜 구획 내의 낮은 pH에 의한 절단에 감수성이거나 또는 프로테아제, 예컨대 종양 조직에서 우선적으로 발현되는 프로테아제, 예컨대 카텝신 (예를 들어, 카텝신 B, C, D)에 의한 절단에 감수성인 링커가 선택될 수 있다.
세포독소의 종류, 치료제를 항체에 접합시키기 위한 링커 및 방법에 대한 추가의 논의에 대해서는 또한 문헌 [Saito et al., (2003) Adv. Drug Deliv. Rev. 55:199-215]; [Trail et al., (2003) Cancer Immunol. Immunother. 52:328-337]; [Payne, (2003) Cancer Cell 3:207-212]; [Allen, (2002) Nat. Rev. Cancer 2:750-763]; [Pastan and Kreitman, (2002) Curr. Opin. Investig. Drugs 3:1089-1091]; [Senter and Springer, (2001) Adv. Drug Deliv. Rev. 53:247-264]을 참조한다.
본 발명의 항체는 또한 방사성 면역접합체로도 언급되는 세포독성 방사성 제약물질을 생성하기 위해 방사성 동위원소에 접합될 수 있다. 진단 또는 치료 목적으로 사용하기 위해 항체에 접합될 수 있는 방사성 동위원소의 예는 아이오딘131, 인듐111, 이트륨9 0, 및 루테튬177을 포함하고 이로 제한되지 않는다. 방사성 면역접합체를 제조하는 방법은 당업계에 확립되어 있다. 제발린TM (DEC 파마슈티칼스 (DEC Pharmaceuticals)) 및 벡사 (Bexxar)TM (코릭사 파마슈티칼스 (Corixa Pharmaceuticals))를 비롯한 방사성 면역접합체의 예는 상업상 이용가능하고, 본 발명의 항체를 사용하여 방사성 면역접합체를 제조하기 위해 유사한 방법을 사용할 수 있다. 특정 실시양태에서, 대환식 (macrocyclic) 킬레이터는 링커 분자를 통해 항체에 부착될 수 있는 1,4,7,10-테트라아자시클로도데칸-N,N',N",N'"-테트라아세트산 (DOTA)이다. 상기 링커 분자는 당업계에 일반적으로 공지되어 있고, 각각 그 전부가 참고로 포함된 문헌 [Denardo et al., (1998) Clin Cancer Res. 4(10):2483-90]; [Peterson et al., (1999) Bioconjug. Chem. 10(4):553-7]; 및 [Zimmerman et al., (1999) Nucl. Med. Biol. 26(8):943-50]에 기재되어 있다.
치료 모이어티를 항체에 접합시키기 위한 기술은 공지되어 있다. 예를 들어, 문헌 [Arnon et al., "Monoclonal Antibodies For Immunotargeting Of Drugs In Cancer Therapy", in Monoclonal Antibodies And Cancer Therapy, Reisfeld et al. (eds.), pp. 243- 56 (Alan R. Liss, Inc. 1985)]; [Hellstrom et al., "Antibodies For Drug Delivery", in Controlled Drug Delivery (2nd Ed.), Robinson et al. (eds.), pp. 623-53 (Marcel Dekker, Inc. 1987)]; [Thorpe, "Antibody Carriers Of Cytotoxic Agents In Cancer Therapy: A Review", in Monoclonal Antibodies 84: Biological And Clinical Applications, Pinchera et al. (eds.), pp. 475-506 (1985)]; ["Analysis, Results, And Future Prospective Of The Therapeutic Use Of Radiolabeled Antibody In Cancer Therapy", in Monoclonal Antibodies For Cancer Detection And Therapy, Baldwin et al. (eds.), pp. 303-16 (Academic Press 1985)], 및 [Thorpe et al., (1982) Immunol. Rev. 62:119-58]을 참조한다.
항체는 또한 표적 항원의 면역검정 또는 정제에 특히 유용한 고체 지지체에 부착될 수 있다. 상기 고체 지지체는 유리, 셀룰로스, 폴리아크릴아미드, 나일론, 폴리스티렌, 폴리비닐 클로라이드 또는 폴리프로필렌을 포함하고 이로 제한되지 않는다.
본 발명의 항체의 생산 방법
(i) 항체를 코딩하는 핵산
본 발명은 상기한 LRP6 항체 사슬의 절편 또는 도메인을 포함하는 폴리펩티드를 코딩하는 실질적으로 정제된 핵산 분자를 제공한다. 본 발명의 핵산의 일부는 서열 14, 34, 및 60에 제시된 프로펠러 1 항체 중쇄 가변 영역을 코딩하는 뉴클레오티드 서열, 및/또는 서열 13, 33, 및 59에 제시된 경쇄 가변 영역을 코딩하는 뉴클레오티드 서열을 포함한다. 특정 실시양태에서, 핵산 분자는 표 1에서 확인되는 것이다. 본 발명의 핵산의 일부는 82, 106, 및 128에 제시된 프로펠러 3 항체 중쇄 가변 영역을 코딩하는 뉴클레오티드 서열, 및/또는 서열 81, 105, 및 127에 제시된 경쇄 가변 영역을 코딩하는 뉴클레오티드 서열을 포함한다. 본 발명의 일부 다른 핵산 분자는 표 1에서 확인되는 것의 뉴클레오티드 서열에 실질적으로 동일한 (예를 들어, 적어도 65%, 80%, 95%, 또는 99%) 뉴클레오티드 서열을 포함한다. 적절한 발현 벡터로부터 발현될 때, 상기 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드는 LRP6 항원 결합 능력을 보일 수 있다.
또한, 본 발명에서는 상기 제시된 LRP6 항체의 중쇄 또는 경쇄로부터의 적어도 1개의 CDR 영역 및 일반적으로는 3개의 모든 CDR 영역을 코딩하는 폴리뉴클레오티드가 제공된다. 일부 다른 폴리뉴클레오티드는 상기 제시된 LRP6 항체의 중쇄 및/또는 경쇄의 가변 영역 서열을 모두 또는 실질적으로 모두 코딩한다. 코드의 축퇴성으로 인해, 다양한 핵산 서열이 각각의 이뮤노글로불린 아미노산 서열을 코딩할 것이다.
본 발명의 핵산 분자는 항체의 가변 영역 및 불변 영역 둘 모두를 코딩할 수 있다. 본 발명의 핵산 서열의 일부는 서열 14, 34, 및 60에 제시된 프로펠러 1 항체의 성숙 중쇄 가변 영역 서열에 실질적으로 동일한 (예를 들어, 적어도 80%, 90%, 또는 99%) 성숙 중쇄 가변 영역 서열을 코딩하는 뉴클레오티드를 포함한다. 일부 다른 핵산 서열은 서열 13, 33, 및 59에 제시된 프로펠러 1 항체의 성숙 경쇄 가변 영역 서열에 실질적으로 동일한 (예를 들어, 적어도 80%, 90%, 또는 99%) 성숙 경쇄 가변 영역 서열을 코딩하는 뉴클레오티드를 포함한다.
본 발명의 핵산 분자는 항체의 가변 영역 및 불변 영역 둘 모두를 코딩할 수 있다. 본 발명의 핵산 서열의 일부는 서열 82, 106, 및 128에 제시된 프로펠러 3 항체의 성숙 중쇄 가변 영역 서열에 실질적으로 동일한 (예를 들어, 적어도 80%, 90%, 또는 99%) 성숙 중쇄 가변 영역 서열을 코딩하는 뉴클레오티드를 포함한다. 일부 다른 핵산 서열은 서열 81, 105, 및 127에 제시된 프로펠러 3 항체의 성숙 경쇄 가변 영역 서열에 실질적으로 동일한 (예를 들어, 적어도 80%, 90%, 또는 99%) 성숙 경쇄 가변 영역 서열을 코딩하는 뉴클레오티드를 포함한다.
폴리뉴클레오티드 서열은 LRP6 항체 또는 그의 결합 단편을 코딩하는 기존의 서열 (예를 들어, 하기 실시예에 기재된 바와 같은 서열)의 드 노보 (de novo) 고체상 DNA 합성 또는 PCR 돌연변이 유발에 의해 생성될 수 있다. 핵산의 직접적인 화학적 합성은 당업계에 공지된 방법, 예컨대 문헌 [Narang et al., (1979), Meth. Enzymol. 68:90]의 포스포트리에스테르 방법, 문헌 [Brown et al., (1979) Meth. Enzymol. 68:109]의 포스포디에스테르 방법, 문헌 [Beaucage et al., (1981) Tetra. Lett., 22:1859]의 디에틸포스포르아미다이트 방법, 및 미국 특허 번호 4,458,066의 고체 지지체 방법으로 달성될 수 있다. PCR에 의해 폴리뉴클레오티드 서열에 돌연변이를 도입하는 것은 예를 들어 문헌 [PCR Technology: Principles and Applications for DNA Amplification, H.A. Erlich (Ed.), Freeman Press, NY, NY, 1992], [PCR Protocols: A Guide to Methods and Applications, Innis et al. (Ed.), Academic Press, San Diego, CA, 1990], [Mattila et al., (1991) Nucleic Acids Res. 19:967] 및 [Eckert et al., (1991) PCR Methods and Applications 1:17]에 기재된 바와 같이 하여 수행될 수 있다.
또한, 본 발명에서는 상기 기재된 LRP6 항체를 생성하기 위한 발현 벡터 및 숙주 세포가 제공된다. LRP6 항체 사슬 또는 결합 단편을 코딩하는 폴리뉴클레오티드를 발현시키기 위해 다양한 발현 벡터를 사용할 수 있다. 포유동물 숙주 세포에서 항체를 생성하기 위해 바이러스-기반 및 비-바이러스 발현 벡터 둘 모두가 사용될 수 있다. 비-바이러스 벡터 및 시스템은 플라스미드, 에피솜 벡터 (일반적으로, 단백질 또는 RNA 발현을 위한 발현 카세트를 가짐), 및 인간 인공 염색체 (예를 들어, 문헌 [Harrington et al., (1997) Nat Genet 15:345] 참조)를 포함한다. 예를 들어, 포유동물 (예를 들어, 인간) 세포에서 LRP6 폴리뉴클레오티드 및 폴리펩티드의 발현에 유용한 비-바이러스 벡터는 pThioHis A, B 및 C, pcDNA3.1/His, pEBVHis A, B 및 C (인비트로젠 (Invitrogen), 미국 캘리포니아주 샌디에고), MPSV 벡터, 및 다른 단백질을 발현하기 위해 당업계에 공지된 많은 다른 벡터를 포함한다. 유용한 바이러스 벡터는 레트로바이러스, 아데노바이러스, 아데노-연관 바이러스, 헤르페스 바이러스를 기반으로 하는 벡터, SV40, 유두종 바이러스, HBP 엡스타인 바 (Epstein Barr) 바이러스를 기반으로 하는 벡터, 우두 바이러스 벡터 및 셈리키 포레스트 (Semliki Forest) 바이러스 (SFV)를 기반으로 하는 벡터를 포함한다. 문헌 [Brent et al., (1995), 상기 문헌]; [Smith, Annu. Rev. Microbiol. 49:807]; 및 [Rosenfeld et al., (1992) Cell 68:143]을 참조한다.
발현 벡터의 선택은 벡터가 발현되는 의도된 숙주 세포에 따라 달라진다. 일반적으로, 발현 벡터는 LRP6 항체 사슬 또는 단편을 코딩하는 폴리뉴클레오티드에 작동가능하게 연결된 프로모터 및 다른 조절 서열 (예를 들어, 인핸서)을 포함한다. 일부 실시양태에서, 유도가능 프로모터는 유도 조건 하인 경우 이외에는 삽입된 서열의 발현을 저해하기 위해 사용된다. 유도가능 프로모터는 예를 들어 아라비노스, lacZ, 메탈로티오네인 프로모터 또는 열 충격 프로모터를 포함한다. 형질전환된 유기체의 배양물은, 발현 생성물이 숙주 세포에 의해 보다 양호하게 허용되는 서열을 코딩하는 집단으로 편향되지 않으면서 비-유도 조건하에서 증식될 수 있다. 프로모터에 추가하여, LRP6 항체 사슬 또는 단편의 효율적인 발현을 위해 다른 조절 요소가 또한 필요하거나 요구될 수 있다. 이들 요소는 일반적으로 ATG 개시 코돈 및 인접한 리보좀 결합 부위 또는 다른 서열을 포함한다. 또한, 발현 효율은 사용하는 세포 시스템에 적절한 인핸서를 포함시켜 향상될 수 있다 (예를 들어, 문헌 [Scharf et al., (1994) Results Probl. Cell Differ. 20:125] 및 [Bittner et al., (1987) Meth. Enzymol., 153:516] 참조). 예를 들어, SV40 인핸서 또는 CMV 인핸서가 포유동물 숙주 세포에서의 발현 증가를 위해 사용될 수 있다.
발현 벡터는 또한 삽입된 LRP6 항체 서열에 의해 코딩되는 폴리펩티드와 융합 단백질을 형성하는 분비 신호 서열 위치를 제공할 수 있다. 보다 흔히, 삽입된 LRP6 항체 서열을 신호 서열에 연결한 후에 벡터에 포함시킨다. LRP6 항체 경쇄 및 중쇄 가변 도메인을 코딩하는 서열을 수용하기 위해 사용되는 벡터는 때로 불변 영역 또는 그의 일부를 또한 코딩한다. 이러한 벡터는 불변 영역과의 융합 단백질로서의 가변 영역의 발현을 허용함으로써 무손상 항체 또는 그의 단편을 생성할 수 있다. 일반적으로, 이러한 불변 영역은 인간의 것이다.
LRP6 항체 사슬을 보유하고 발현하는 숙주 세포는 원핵 또는 진핵 세포일 수 있다. 이. 콜라이는 본 발명의 폴리뉴클레오티드를 클로닝하고 발현시키는데 유용한 원핵 숙주 중 하나이다. 사용하기 적합한 다른 미생물 숙주는 바실러스, 예를 들어 바실러스 섭틸리스 (Bacillus subtilis), 및 다른 장내세균, 예를 들어 살모넬라 (Salmonella), 세라티아 (Serratia) 및 다양한 슈도모나스 종을 포함한다. 이들 원핵 숙주에서, 일반적으로 숙주 세포와 상용성인 발현 제어 서열 (예를 들어, 복제 기점)을 함유하는 발현 벡터를 또한 제조할 수 있다. 또한, 임의의 수의 다양한 공지된 프로모터, 예를 들어 락토스 프로모터 시스템, 트립토판 (trp) 프로모터 시스템, 베타-락타마제 프로모터 시스템, 또는 파지 람다로부터의 프로모터 시스템이 존재할 것이다. 프로모터는 일반적으로 임의로는 오퍼레이터 서열과 함께 발현을 제어하고, 전사 및 번역을 개시하고 종료하기 위한 리보좀 결합 부위 서열 등을 갖는다. 본 발명의 LRP6 폴리펩티드를 발현시키기 위해 다른 미생물, 예를 들어 효모가 또한 사용될 수 있다. 바큘로바이러스 벡터와 조합된 곤충 세포도 사용될 수 있다.
일부 바람직한 실시양태에서, 포유동물 숙주 세포가 본 발명의 LRP6 항체를 발현시키고 제조하는데 사용된다. 예를 들어, 이것들은 내인성 이뮤노글로불린 유전자를 발현하는 하이브리도마 세포주 (예를 들어, 실시예에서 설명되는 바와 같은 1D6.C9 골수종 하이브리도마 클론) 또는 외인성 발현 벡터를 보유하는 포유동물 세포주 (예를 들어, 아래에서 예시되는 SP2/0 골수종 세포)일 수 있다. 이것들은 임의의 정상적인 사망할 동물 또는 인간 세포, 또는 정상적이거나 비정상적인 불멸 동물 또는 인간 세포를 포함한다. 예를 들어, CHO 세포주, 다양한 Cos 세포주, HeLa 세포, 골수종 세포주, 형질전환된 B-세포 및 하이브리도마를 비롯하여 무손상 이뮤노글로불린을 분비할 수 있는 많은 적합한 숙주 세포주가 개발된 바 있다. 포유동물 조직 세포 배양을 이용하여 폴리펩티드를 발현시키는 것은 예를 들어 문헌 [Winnacker, FROM GENES TO CLONES, VCH Publishers, N.Y., N.Y., 1987]에서 일반적으로 논의된다. 포유동물 숙주 세포를 위한 발현 벡터는 발현 제어 서열, 예컨대 복제 기점, 프로모터 및 인핸서 (예를 들어, 문헌 [Queen, et al., (1986) Immunol. Rev. 89:49-68] 참조), 및 필요한 프로세싱 정보 부위, 예컨대 리보좀 결합 부위, RNA 스플라이스 부위, 폴리아데닐화 부위 및 전사 종결자 서열을 포함할 수 있다. 이들 발현 벡터는 대체로 포유동물 유전자 또는 포유동물 바이러스로부터 유래된 프로모터를 함유한다. 적합한 프로모터는 구성적, 세포 유형-특이적, 단계-특이적 및/또는 조정가능하거나 조절가능한 것일 수 있다. 유용한 프로모터는 메탈로티오네인 프로모터, 구성적 아데노바이러스 주요 후기 프로모터, 덱사메타손-유도가능 MMTV 프로모터, SV40 프로모터, MRP polIII 프로모터, 구성적 MPSV 프로모터, 테트라사이클린-유도가능 CMV 프로모터 (예컨대, 인간 최조기 CMV 프로모터), 구성적 CMV 프로모터, 및 당업계에 공지된 프로모터-인핸서 조합물을 포함하고, 이로 제한되지 않는다.
관심있는 폴리뉴클레오티드 서열을 함유하는 발현 벡터를 도입하는 방법은 세포 숙주의 유형에 따라 달라진다. 예를 들어, 염화칼슘 형질감염은 통상적으로 원핵 세포에 대해 사용되고, 인산칼슘 처리 또는 전기천공은 다른 세포 숙주에 대해 사용될 수 있다 (일반적으로, 문헌 [Sambrook, et al., 상기 문헌] 참조). 다른 방법은 예를 들어 전기천공, 인산칼슘 처리, 리포솜-매개 형질전환, 주사 및 미세주사, 발리스틱 (ballistic) 방법, 비로좀, 면역리포솜, 다중양이온:핵산 접합체, 네이키드 DNA, 인공 비리온, 헤르페스 바이러스 구조 단백질 VP22에 대한 융합 ([Elliot and O'Hare, (1997) Cell 88:223]), DNA 흡수 증진제, 및 생체외 형질도입을 포함한다. 재조합 단백질의 장기간 고수율 생성을 위해, 종종 안정적인 발현이 요구될 것이다. 예를 들어, LRP6 항체 사슬 또는 결합 단편을 안정적으로 발현하는 세포주는 바이러스 복제 기점 또는 내인성 발현 요소 및 선택가능한 마커 유전자를 함유하는 본 발명의 발현 벡터를 사용하여 제조할 수 있다. 벡터의 도입 후에 세포를 1일 내지 2일 동안 영양강화 배지에서 성장시킨 후, 선택 배지로 전환할 수 있다. 선택가능한 마커의 목적은 선택에 대한 내성을 부여하기 위한 것이고, 이의 존재는 도입된 서열을 성공적으로 발현하는 세포가 선택 배지에서 성장하게 한다. 안정적으로 형질감염된 내성 세포는 세포 유형에 적절한 조직 배양 기술을 이용하여 증식될 수 있다.
(
ii
)
모노클로날
항체의 생성
모노클로날 항체 (mAb)는 통상적인 모노클로날 항체 방법, 예를 들어 문헌 [Kohler and Milstein, (1975) Nature 256:495]의 표준 체세포 혼성화 기술을 비롯한 다양한 기술에 의해 생산될 수 있다. 모노클로날 항체를 생성하는 많은 기술, 예를 들어 B 림프구의 바이러스성 또는 발암성 형질전환을 이용할 수 있다.
하이브리도마를 제조하기 위한 동물 시스템은 뮤린 시스템이다. 마우스에서 하이브리도마 생산은 잘 확립된 절차이다. 면역화 프로토콜 및 융합을 위한 면역화된 비장세포의 단리를 위한 기술은 당업계에 공지되어 있다. 융합 파트너 (예를 들어, 뮤린 골수종 세포) 및 융합 절차도 공지되어 있다.
본 발명의 키메라 또는 인간화 항체는 상기 기재된 바와 같이 제조된 뮤린 모노클로날 항체의 서열을 기초로 하여 제조할 수 있다. 표준 분자 생물학 기술을 이용하여, 중쇄 및 경쇄 이뮤노글로불린을 코딩하는 DNA를 관심있는 뮤린 하이브리도마로부터 얻고 이것을 조작하여 비-뮤린 (예를 들어, 인간) 이뮤노글로불린 서열을 함유하게 할 수 있다. 예를 들어, 키메라 항체를 생성하기 위해서, 당업계에 공지된 방법을 이용하여 뮤린 가변 영역을 인간 불변 영역에 연결할 수 있다 (예를 들어, 미국 특허 번호 4,816,567 (Cabilly et al.) 참조). 인간화 항체를 생성하기 위해서, 뮤린 CDR 영역을 당업계에 공지된 방법을 이용하여 인간 프레임워크 내로 삽입할 수 있다. 예를 들어, 미국 특허 번호 5225539 (윈터), 및 미국 특허 번호 5530101; 5585089; 5693762 및 6180370 (퀸 등)을 참조한다.
특정 실시양태에서, 본 발명의 항체는 인간 모노클로날 항체이다. LRP6에 대한 이러한 인간 모노클로날 항체는 마우스 면역계가 아닌 인간 면역계의 일부를 보유하는 트랜스제닉 또는 트랜스크로모조멀 마우스를 사용하여 생성시킬 수 있다. 이들 트랜스제닉 및 트랜스크로모조멀 마우스는 본원에서 각각 HuMAb 마우스 및 KM 마우스로 지칭되는 마우스를 포함하고, 본원에서는 이들을 "인간 Ig 마우스"라고 통칭한다.
HuMAb 마우스® (메다렉스, 인크. (Medarex, Inc.))는 내인성 μ 및 κ 사슬 로커스를 불활성화시키는 표적화 돌연변이와 함께, 재배열되지 않는 인간 중쇄 (μ 및 γ) 및 κ 경쇄 이뮤노글로불린 서열을 코딩하는 인간 이뮤노글로불린 유전자 미니로커스 (miniloci)를 함유한다 (예를 들어, 문헌 [Lonberg, et al., (1994) Nature 368(6474): 856-859] 참조). 따라서, 상기 마우스는 마우스 IgM 또는 κ의 발현 감소를 나타내고, 면역화에 반응하여, 도입된 인간 중쇄 및 경쇄 트랜스진에서 클래스 스위칭 및 체세포 돌연변이가 일어나서 고친화도 인간 IgGκ 모노클로날을 생성한다 ([Lonberg et al., 1994 상기 문헌], 문헌 [Lonberg, (1994) Handbook of Experimental Pharmacology 113:49-101]에서 검토됨, [Lonberg and Huszar, (1995) Intern. Rev. Immunol.13: 65-93], 및 [Harding and Lonberg, (1995) Ann. N. Y. Acad. Sci. 764:536-546]). HuMAb 마우스의 제조 및 사용, 및 이러한 마우스에 의해 보유되는 게놈 변형은 그 모든 내용이 본원에 참고로 구체적으로 포함되는 하기 문헌에 추가로 기재되어 있다: [Taylor et al., (1992) Nucleic Acids Research 20:6287-6295]; [Chen et al., (1993) International Immunology 5:647-656]; [Tuaillon et al., (1993) Proc. Natl. Acad. Sci. USA 94:3720-3724]; [Choi et al., (1993) Nature Genetics 4:117-123]; [Chen et al., (1993) EMBO J. 12:821-830]; [Tuaillon et al., (1994) J. Immunol. 152:2912-2920]; [Taylor et al., (1994) International Immunology 579-591]; 및 [Fishwild et al., (1996) Nature Biotechnology 14:845-851]. 추가로, 미국 특허 번호 5,545,806; 5,569,825; 5,625,126; 5,633,425; 5,789,650; 5,877,397; 5,661,016; 5,814,318; 5,874,299; 및 5,770,429 (론버그(Lonberg) 및 케이(Kay) (상기 특허 전부)); 미국 특허 번호 5,545,807 (수라니(Surani) 등); PCT 공개 번호 WO 92103918, WO 93/12227, WO 94/25585, WO 97/13852, WO 98/24884 및 WO 99/45962 (론버그 및 케이 (상기 공개 전부)); 및 PCT 공개 번호 WO 01/14424 (코르만(Korman) 등)를 참조한다.
또 다른 실시양태에서, 본 발명의 인간 항체는 트랜스진 및 도입염색체 (transchromosome) 상에 인간 이뮤노글로불린 서열을 보유하는 마우스, 예를 들어 인간 중쇄 트랜스진 및 인간 경쇄 도입염색체를 보유하는 마우스를 사용하여 생성할 수 있다. 이러한 마우스는 본원에서 "KM 마우스"라 지칭되며, PCT 공개 WO 02/43478 (이시다(Ishida) 등)에 상세하게 기재되어 있다.
추가로, 인간 이뮤노글로불린 유전자를 발현하는 대안적인 트랜스제닉 동물 시스템이 당업계에서 이용가능하고, 본 발명의 LRP6 항체를 생성하는데 사용될 수 있다. 예를 들어, 제노마우스 (Xenomouse) (압제닉스, 인크. (Abgenix, Inc.))로 지칭되는 대안적인 트랜스제닉 시스템을 사용할 수 있다. 이러한 마우스는 예를 들어 미국 특허 번호 5,939,598; 6,075,181; 6,114,598; 6,150,584 및 6,162,963 (쿠체라파티(Kucherlapati) 등)에 기재되어 있다.
추가로, 인간 이뮤노글로불린 유전자를 발현하는 대안적인 트랜스크로모조멀 동물 시스템이 당업계에서 이용가능하고, 본 발명의 LRP6 항체를 생성하는데 사용될 수 있다. 예를 들어, "TC 마우스"로 지칭되는, 인간 중쇄 도입염색체 및 인간 경쇄 도입염색체 둘 모두를 보유하는 마우스를 사용할 수 있고, 이러한 마우스는 문헌 [Tomizuka et al., (2000) Proc. Natl. Acad. Sci. USA 97:722-727]에 기재되어 있다. 추가로, 인간 중쇄 및 경쇄 도입색체를 보유하는 소가 당업계에서 설명된 바 있고 ([Kuroiwa et al., (2002) Nature Biotechnology 20:889-894]), 본 발명의 LRP6 항체를 생성하는데 사용될 수 있다.
본 발명의 인간 모노클로날 항체는 인간 이뮤노글로불린 유전자의 라이브러리를 스크리닝하기 위한 파지 디스플레이 방법을 이용하여 제조할 수도 있다. 인간 항체를 단리하기 위한 이러한 파지 디스플레이 방법은 당업계에서 확립되어 있거나 하기 실시예에서 설명된다. 예를 들어: 미국 특허 번호 5,223,409; 5,403,484; 및 5,571,698 (라드너(Ladner) 등); 미국 특허 번호 5,427,908 및 5,580,717 (다우어(Dower) 등); 미국 특허 번호 5,969,108 및 6,172,197 (맥카퍼티(McCafferty) 등); 및 미국 특허 번호 5,885,793; 6,521,404; 6,544,731; 6,555,313; 6,582,915 및 6,593,081 (그리피스(Griffiths) 등)을 참조한다.
또한, 본 발명의 인간 모노클로날 항체는 면역화시에 인간 항체 반응이 발생할 수 있도록 인간 면역 세포가 재구성된 SCID 마우스를 사용하여 제조할 수 있다. 이러한 마우스는 예를 들어 미국 특허 번호 5,476,996 및 5,698,767 (윌슨(Wilson) 등)에 기재되어 있다.
(
iii
)
프레임워크
또는
Fc
조작
본 발명의 조작된 항체는 예를 들어 항체의 특성을 개선하기 위해 VH 및/또는 VL 내의 프레임워크 잔기에 대해 변형이 이루어진 것을 포함한다. 일반적으로, 이러한 프레임워크 변형은 항체의 면역원성을 감소시키기 위해 이루어진다. 예를 들어, 한 방법은 1개 이상의 프레임워크 잔기를 상응하는 배선 서열로 "역돌연변이"시키는 것이다. 보다 구체적으로, 체세포 돌연변이가 일어난 항체는 이러한 항체가 유래된 배선 서열과는 상이한 프레임워크 잔기를 함유할 수 있다. 이러한 잔기는 항체 프레임워크 서열을 항체가 유래된 배선 서열과 비교함으로써 확인할 수 있다. 프레임워크 영역 서열을 그의 배선 입체형태로 회복하기 위해, 예를 들어 부위-지정 돌연변이 유발에 의해 체세포 돌연변이를 배선 서열로 "역돌연변이"시킬 수 있다. 이러한 "역돌연변이" 항체 역시 본 발명에 포함되는 것으로 의도된다.
또 다른 유형의 프레임워크 변형은 프레임워크 영역 내 또는 심지어는 1개 이상의 CDR 영역 내의 1개 이상의 잔기를 돌연변이시켜 T 세포-에피토프를 제거함으로써 항체의 잠재적 면역원성을 감소시키는 것을 포함한다. 이러한 방법은 "탈면역화"로도 지칭되고, 미국 특허 공개 번호 20030153043 (카(Carr) 등)에 추가로 상세하게 기재되어 있다.
프레임워크 또는 CDR 영역 내에서 이루어진 변형에 추가하여 또는 이러한 변형에 대한 대안으로, 본 발명의 항체는 일반적으로 항체의 하나 이상의 기능적 특성, 예컨대 혈청 반감기, 보체 고정, Fc 수용체 결합, 및/또는 항원-의존적 세포성 세포독성을 변경시키는 변형을 Fc 영역 내에 포함하도록 조작될 수 있다. 추가로, 본 발명의 항체를 화학적으로 변형시키거나 (예를 들어, 1개 이상의 화학적 모이어티를 항체에 부착시킬 수 있음), 또는 그의 글리코실화를 변경시켜 항체의 하나 이상의 기능적 특성이 다시 변경되도록 변형시킬 수도 있다. 이러한 실시양태는 각각 하기에서 추가로 상세하게 설명된다. Fc 영역 내 잔기의 넘버링은 [Kabat (상기 문헌)]에 따른 것이다.
한 실시양태에서, CH1의 힌지 영역을 변형시켜 힌지 영역 내의 시스테인 잔기의 수를 변경, 예를 들어 증가 또는 감소시킨다. 이러한 방법은 미국 특허 번호 5,677,425 (보드머(Bodmer) 등)에 추가로 기재되어 있다. CH1의 힌지 영역 내의 시스테인 잔기 수는 예를 들어 경쇄 및 중쇄의 조립을 용이하게 하거나 항체의 안정성을 증가 또는 감소시키도록 변경된다.
또 다른 실시양태에서, 항체의 Fc 힌지 영역은 항체의 생물학적 반감기를 감소시키기 위해 돌연변이된다. 보다 구체적으로, 항체가 천연 Fc-힌지 도메인 SpA 결합에 비해 손상된 스타필로코쿠스 단백질 A (SpA) 결합을 갖도록 하나 이상의 아미노산 돌연변이가 Fc-힌지 단편의 CH2-CH3 도메인 계면 영역 내로 도입된다. 상기 방법은 미국 특허 번호 6,165,745 (워드(Ward) 등)에 추가로 상세히 설명되어 있다.
또 다른 실시양태에서, Fc 영역은 항체의 이펙터 기능을 변경하기 위해 1개 이상의 아미노산 잔기를 상이한 아미노산 잔기로 대체시킴으로써 변경된다. 예를 들어, 1개 이상의 아미노산을 상이한 아미노산 잔기로 대체하여 항체가 이펙터 리간드에 대해 변경된 친화도를 갖지만 모 항체의 항원-결합 능력은 보유하게 할 수 있다. 친화도가 변경된 이펙터 리간드는 예를 들어 Fc 수용체 또는 보체의 C1 성분일 수 있다. 상기 방안은 미국 특허 번호 5,624,821 및 5,648,260 (둘 모두 윈터 등)에 추가로 상세히 설명되어 있다
또 다른 실시양태에서, 아미노산 잔기로부터 선택된 1개 이상의 아미노산을 상이한 아미노산 잔기로 대체시켜 항체가, 변경된 C1q 결합 및/또는 감소되거나 파괴된 보체 의존적 세포독성 (CDC)을 갖도록 할 수 있다. 이러한 방법은 미국 특허 번호 6,194,551 (이두소기(Idusogie) 등)에 추가로 상세하게 기재되어 있다.
또 다른 실시양태에서, 1개 이상의 아미노산 잔기를 변경시켜서 보체를 고정하는 항체의 능력을 변경할 수 있다. 이러한 방법은 PCT 공개 WO 94/29351 (보드머 등)에 추가로 상세하게 기재되어 있다.
또 다른 실시양태에서, Fc 영역은 1개 이상의 아미노산을 변형시킴으로써 항체 의존적 세포성 세포독성 (ADCC)을 매개하는 항체의 능력이 증가되고/되거나 Fcγ 수용체에 대한 항체의 친화도가 증가되도록 변형된다. 이러한 방법은 PCT 공개 WO 00/42072 (프레스타)에 추가로 상세하게 설명되어 있다. 추가로, 인간 IgG1에서 FcγRI, FcγRII, FcγRIII 및 FcRn에 대한 결합 부위가 맵핑되었고, 결합이 개선된 변이체가 문헌에 기재된 바 있다 (문헌 [Shields et al., (2001) J. Biol. Chen. 276:6591-6604] 참조).
또 다른 실시양태에서, 항체의 글리코실화가 변형된다. 예를 들어, 탈글리코실화 항체가 생성될 수 있다 (즉, 항체에 글리코실화가 결여됨). 글리코실화는 예를 들어 "항원"에 대한 항체의 친화도가 증가되도록 변형시킬 수 있다. 이러한 탄수화물 변형은 예를 들어 항체 서열 내의 1개 이상의 글리코실화 부위를 변경시켜 달성될 수 있다. 예를 들어, 1개 이상의 가변 영역 프레임워크 글리코실화 부위를 제거하여 그 부위에서 글리코실화가 일어나지 않도록 하는 1개 이상의 아미노산 치환이 일어날 수 있다. 이러한 탈글리코실화는 항원에 대한 항체의 친화도를 증가시킬 수 있다. 이러한 방법은 미국 특허 번호 5,714,350 및 6,350,861 (코(Co) 등)에 더욱 상세하게 기재되어 있다.
추가로 또는 별법으로, 변경된 유형의 글리코실화를 갖는 항체, 예컨대 푸코실 잔기 수가 감소된 저푸코실화 (hypofucosylation) 항체 또는 이분화 GlcNac 구조가 증가된 항체가 생성될 수 있다. 이러한 변경된 글리코실화 패턴이 항체의 ADCC 능력을 증가시키는 것으로 입증된 바 있다. 이러한 탄수화물 변형은 예를 들어 변경된 글리코실화 기구를 갖는 숙주 세포 내에서 항체를 발현시켜서 달성될 수 있다. 변경된 글리코실화 기구를 갖는 세포는 당업계에 기재되어 있으며, 본 발명의 재조합 항체를 발현시켜 변경된 글리코실화를 갖는 항체를 생성하는 숙주 세포로서 사용될 수 있다. 예를 들어, EP 1,176,195 (행(Hang) 등)에는 세포주에서 발현된 항체가 저푸코실화를 보이도록 푸코실 트랜스퍼라제를 코딩하는 FUT8 유전자가 기능적으로 파괴된 세포주를 기재되어 있다. PCT 공개 WO 03/035835 (프레스타)는 Asn(297)-연결된 탄수화물에 푸코스를 부착시키는 능력이 감소되어 숙주 세포 내에서 발현된 항체에서 저푸코실화가 일어나는 변이체 CHO 세포주인 Lecl3 세포를 기재하고 있다 (또한, 문헌 [Shields et al., (2002) J. Biol. Chem. 277:26733-26740] 참조). PCT 공개 WO 99/54342 (우마나(Umana) 등)는 당단백질-변형 글리코실 트랜스퍼라제 (예를 들어, 베타(1,4)-N-아세틸글루코스아미닐트랜스퍼라제 III (GnTIII))를 발현하도록 조작된 세포주를 기재하며, 이와 같이 조작된 세포주에서 발현된 항체는 증가된 이분화 GlcNac 구조를 보여 항체의 ADCC 활성을 증가시킨다 (또한 문헌 [Umana et al., (1999) Nat. Biotech. 17:176-180] 참조).
또 다른 실시양태에서, 항체는 그의 생물학적 반감기를 증가시키기 위해 변형된다. 다양한 방법이 가능하다. 예를 들어, 미국 특허 번호 6,277,375 (워드)에 기재된 바와 같이, 돌연변이 T252L, T254S, T256F 중 1종 이상이 도입될 수 있다. 별법으로, 미국 특허 번호 5,869,046 및 6,121,022 (프레스타 등)에 기재된 바와 같이, 생물학적 반감기를 증가시키기 위해서 IgG의 Fc 영역의 CH2 도메인의 2개 루프로부터의 샐비지 수용체 결합 에피토프를 함유하도록 항체를 CH1 또는 CL 영역 내에서 변경시킬 수 있다.
(
iv
) 변경된 항체의 조작 방법
상기 논의된 바와 같이, 본원에서 제시된 VH 및 VL 서열 또는 전장 중쇄 및 경쇄 서열을 갖는 LRP6 항체를 사용하여, 전장 중쇄 및/또는 경쇄 서열, VH 및/또는 VL 서열, 또는 그에 부착된 불변 영역(들)을 변형시켜서 새로운 LRP6 항체를 생성할 수 있다. 따라서, 본 발명의 또 다른 측면에서, 본 발명의 LRP6 항체의 구조적 특징을 이용하여 본 발명의 항체의 하나 이상의 기능적 특성, 예컨대 인간 LRP6에 대한 결합 및 또한 LRP6의 하나 이상의 기능적 특성의 억제를 보유하는, 구조적으로 관련된 LRP6 항체를 생성한다.
예를 들어, 본 발명의 항체의 1개 이상의 CDR 영역 또는 그의 돌연변이는 공지의 프레임워크 영역 및/또는 다른 CDR과 재조합 방식으로 조합되어, 상기 논의된 바와 같이 재조합 방식으로 조작된 본 발명의 추가의 LRP6 항체를 생성할 수 있다. 다른 유형의 변형은 상기한 섹션에 기재된 것들을 포함한다. 조작 방법을 위한 출발 물질은 본원에서 제공되는 1종 이상의 VH 및/또는 VL 서열, 또는 그의 1종 이상의 CDR 영역이다. 조작된 항체를 생성하기 위해, 본원에서 제공되는 1종 이상의 VH 및/또는 VL 서열 또는 그의 1종 이상의 CDR 영역을 갖는 항체를 실제로 제조할 (즉, 단백질로서 발현시킬) 필요는 없다. 오히려, 서열(들) 내에 함유된 정보를 출발 물질로서 사용하여 원래의 서열(들)로부터 유래된 "제2 세대" 서열(들)을 생성한 후, 상기 "제2 세대" 서열(들)을 단백질로서 제조 및 발현시킨다.
따라서, 또 다른 실시양태에서, 본 발명은 중쇄 가변 영역 항체 서열 및/또는 경쇄 가변 영역 항체 서열 내의 1개 이상의 아미노산 잔기를 변경시켜 1종 이상의 변경된 항체 서열을 생성하고, 상기 변경된 항체 서열을 단백질로서 발현시키는 것을 포함하는, 서열 1, 21, 및 47로 이루어진 군 중에서 선택된 CDR1 서열, 서열 2, 22, 및 48로 이루어진 군 중에서 선택된 CDR2 서열, 및/또는 서열 3, 23, 및 49로 이루어진 군 중에서 선택된 CDR3 서열을 갖는 중쇄 가변 영역 항체 서열; 및 서열 4, 24, 및 50으로 이루어진 군 중에서 선택된 CDR1 서열, 서열 5, 25, 및 51로 이루어진 군 중에서 선택된 CDR2 서열, 및/또는 서열 6, 26, 및 52로 이루어진 군 중에서 선택된 CDR3 서열을 갖는 경쇄 가변 영역 항체 서열로 이루어지는 프로펠러 1 LRP6 항체의 제조 방법을 제공한다.
따라서, 또 다른 실시양태에서, 본 발명은 중쇄 가변 영역 항체 서열 및/또는 경쇄 가변 영역 항체 서열 내의 1개 이상의 아미노산 잔기를 변경시켜 1종 이상의 변경된 항체 서열을 생성하고, 상기 변경된 항체 서열을 단백질로서 발현시키는 것을 포함하는, 서열 69, 93, 및 115로 이루어진 군 중에서 선택된 CDR1 서열, 서열 70, 94, 및 116으로 이루어진 군 중에서 선택된 CDR2 서열, 및/또는 서열 71, 95, 및 117로 이루어진 군 중에서 선택된 CDR3 서열을 갖는 중쇄 가변 영역 항체 서열; 및 서열 91, 107, 및 118로 이루어진 군 중에서 선택된 CDR1 서열, 서열 73, 97, 및 121로 이루어진 군 중에서 선택된 CDR2 서열, 및/또는 서열 74, 98, 및 120으로 이루어진 군 중에서 선택된 CDR3 서열을 갖는 경쇄 가변 영역 항체 서열로 이루어지는 프로펠러 3 LRP6 항체의 제조 방법을 제공한다.
변경된 항체 서열은 또한 고정된 CDR3 서열 또는 US20050255552에 기재된 바와 같은 최소 필수 결합 결정인자, 및 CDR1 및 CDR2 서열에서의 다양성을 갖는 항체 라이브러리를 스크리닝하여 제조될 수 있다. 스크리닝은 항체 라이브러리로부터 항체를 스크리닝하기에 적절한 임의의 스크리닝 기술, 예를 들어 파지 디스플레이 기술에 따라 수행될 수 있다.
표준 분자 생물학 기술을 이용하여, 변경된 항체 서열을 제조하고 발현시킬 수 있다. 변경된 항체 서열(들)에 의해 코딩되는 항체는 본원에서 설명되는 LRP6 항체의 기능적 특성 중 하나, 일부 또는 모두를 보유하는 것이며, 상기 기능적 특성은 인간 및/또는 시노몰구스 LRP6에 대한 특이적 결합을 포함하고 이에 제한되지 않으며, 상기 항체는 LRP6에 결합하고, Wnt 리포터 유전자 검정에서 정형적 Wnt 신호 전달 활성을 억제함으로써 LRP6 생물학적 활성을 억제한다.
변경된 항체의 기능적 특성은 당업계에 이용가능하고/하거나 본원에 기재된 표준 검정, 예컨대 실시예에 제시되는 것 (예를 들어, ELISA)을 이용하여 평가할 수 있다.
본 발명의 항체의 조작 방법의 특정 실시양태에서, 돌연변이는 LRP6 항체 코딩 서열의 전체 또는 일부를 따라 무작위로 또는 선택적으로 도입될 수 있고, 생성되는 변형된 LRP6 항체는 결합 활성 및/또는 본원에 기재된 바와 같은 다른 기능적 특성에 대해 스크리닝될 수 있다. 돌연변이 방법은 당업계에 기재되어 있다. 예를 들어, PCT 공개 WO 02/092780 (쇼트(Short))은 포화 돌연변이 유발, 합성 라이게이션 조립, 또는 이들의 조합을 이용하여 항체 돌연변이를 생성하고 스크리닝하는 방법을 기재한다. 별법으로, PCT 공개 WO 03/074679 (라자르(Lazar) 등)는 항체의 물리화학적 특성을 최적화하는 전산 스크리닝 방법을 이용한 방법을 설명하고 있다.
본 발명의 항체의 특성 결정
본 발명의 항체는 다양한 기능성 검정에 의해 특성을 결정할 수 있다. 예를 들어, 항체는 본원에서 설명되는 Wnt 유전자 검정에서 정형적 Wnt 신호 전달을 억제함으로써 생물학적 활성을 억제하는 그들의 능력, LRP6 단백질 (예를 들어, 인간 및/또는 시노몰구스 LRP6)에 대한 그들의 친화도, 에피토프 비닝 (binning), 단백질분해에 대한 그들의 저항성, 및 Wnt 경로를 차단하는 그들의 능력을 특징으로 할 수 있다. 또한, 항체는 Wnt 리간드의 존재 하에 Wnt 신호를 강화하는 능력을 특징으로 한다. LRP6-매개 Wnt 신호 전달을 측정하기 위해 다양한 방법을 사용할 수 있다. 예를 들어, Wnt 신호 전달 경로는 (i) β-카테닌의 풍부도 및 위치의 측정; 및 (ii) LRP6 또는 다른 하류 Wnt 신호 전달 단백질 (예를 들어 DVL)의 인산화의 측정, 및 (iii) 특이적 유전자 시그너쳐 (signature) 또는 유전자 표적 (예를 들어 c-myc, 사이클린-D, 액신2)의 측정에 의해 모니터링될 수 있다.
LRP6에 결합하는 항체의 능력은 관심있는 항체를 직접 표지함으로써 검출할 수 있거나, 또는 항체는 비표지되고, 결합은 당업계에 공지된 다양한 샌드위치 검정 포맷을 이용하여 간접적으로 검출될 수 있다.
몇몇 실시양태에서, 본 발명의 LRP6 항체는 LRP6 폴리펩티드에 대한 참조 LRP6 항체의 결합을 차단하거나 이와 경쟁한다. 이들은 상기한 완전 인간 LRP6 항체일 수 있다. 또한, 이들은 참조 항체와 동일한 에피토프에 결합하는 다른 마우스, 키메라 또는 인간화 LRP6 항체일 수 있다. 참조 항체 결합을 차단하거나 이와 경쟁하는 능력은 시험되는 LRP6 항체가 참조 항체에 의해 규정되는 것과 동일하거나 유사한 에피토프에 결합하거나, 또는 참조 LRP6 항체에 의해 결합되는 에피토프와 충분히 가까운 에피토프에 결합함을 나타낸다. 상기 항체는 특히 참조 항체에 대해 확인된 유리한 특성을 공유하는 것으로 보인다. 참조 항체를 차단하거나 이와 경쟁하는 능력은 예를 들어 경쟁 결합 검정에 의해 결정할 수 있다. 경쟁 결합 검정을 이용하여, 시험되는 항체를 공통 항원, 예컨대 LRP6 폴리펩티드에 대한 참조 항체의 특이적 결합을 억제하는 능력에 대해 조사한다. 시험 항체는 과량의 시험 항체가 참조 항체의 결합을 실질적으로 억제할 경우 항원에 대한 특이적 결합을 위해 참조 항체와 경쟁한다. 실질적인 억제는 시험 항체가 참조 항체의 특이적 결합을 대체로 적어도 10%, 25%, 50%, 75%, 또는 90% 감소시킴을 의미한다.
LRP6 항체가 LRP6 단백질에 대한 결합을 위해 참조 LRP6 항체와 경쟁하는 것을 평가하기 위해 사용될 수 있는 많은 경쟁 결합 검정이 공지되어 있다. 이들은 예를 들어 고체상 직접적 또는 간접적 방사성 면역검정 (RIA), 고체상 직접적 또는 간접적 효소 면역검정 (EIA), 샌드위치 경쟁 검정 ([Stahli et al., (1983) Methods in Enzymology 9:242-253] 참조); 고체상 직접적 비오틴-아비딘 EIA ([Kirkland et al., (1986) J. Immunol. 137:3614-3619] 참조); 고체상 직접 표지 검정, 고체상 직접 표지 샌드위치 검정 ([Harlow & Lane, 상기 문헌] 참조); I-125 표지를 사용한 고체상 직접 표지 RIA ([Morel et al., (1988) Molec. Immunol. 25:7-15] 참조); 고체상 직접 비오틴-아비딘 EIA (Cheung et al., (1990) Virology 176:546-552); 및 직접 표지 RIA (Moldenhauer et al., (1990) Scand. J. Immunol. 32:77-82)를 포함한다. 일반적으로, 상기 검정은 고체 표면에 결합된 정제된 항원 또는 비표지된 시험 LRP6 항체 및 표지된 참조 항체를 보유하는 세포의 사용을 수반한다. 경쟁적 억제는 시험 항체의 존재 하에 고체 표면 또는 세포에 결합된 표지의 양을 결정함으로써 측정된다. 대체로, 시험 항체는 과량으로 존재한다. 경쟁 검정에 의해 확인된 항체 (경쟁하는 항체)는 참조 항체와 동일한 에피토프에 결합하는 항체 및 참조 항체에 의해 결합되는 에피토프와 가까운, 입체 장애가 발생하기에 충분히 인접한 에피토프에 결합하는 항체를 포함한다.
선택된 LRP6 모노클로날 항체가 특유의 에피토프에 결합하는지 여부를 결정하기 위해, 각각의 항체를 시판 시약 (예를 들어, 피어스 (미국 일리노이주 록포드)의 시약)을 사용하여 비오티닐화시킬 수 있다. 비표지 모노클로날 항체 및 비오티닐화 모노클로날 항체를 사용한 경쟁 연구는 LRP6 폴리펩티드 코팅된 ELISA 플레이트를 사용하여 수행할 수 있다. 비오티닐화 mAb 결합은 스트렙타비딘-알칼리성 포스파타제 프로브로 검출할 수 있다. 정제된 LRP6 항체의 이소형을 결정하기 위해, 이소형 ELISA를 수행할 수 있다. 예를 들어, 마이크로타이터 플레이트의 웰을 1 ㎍/mL의 항-인간 IgG로 4℃에서 밤새 코팅할 수 있다. 1% BSA로 차단한 후, 플레이트를 1 ㎍/mL 이하의 모노클로날 LRP6 항체 또는 정제된 이소형 대조군과 주변 온도에서 1시간 내지 2시간 동안 반응시킨다. 이어서, 웰을 인간 IgG1 또는 인간 IgM-특이적 알칼리성 포스파타제-접합된 프로브와 반응시킬 수 있다. 이어서, 플레이트를 발색시키고 분석하여 정제된 항체의 이소형을 결정할 수 있다.
LRP6 폴리펩티드를 발현하는 살아있는 세포에 대한 모노클로날 LRP6 항체의 결합을 입증하기 위해, 유동 세포측정법 (flow cytometry)을 이용할 수 있다. 간략하게 설명하면, LRP6를 발현하는 세포주 (표준 성장 조건하에 성장시킴)를 0.1% BSA 및 10% 소 태아 혈청을 함유하는 PBS 중 다양한 농도의 LRP6 항체와 혼합하고, 37℃에서 1시간 동안 인큐베이션할 수 있다. 세척 후, 세포를 1차 항체 염색과 동일한 조건 하에 플루오레세인-표지된 항-인간 IgG 항체와 반응시킨다. 샘플은 단일 세포에 대해 게이팅되는, 광 및 측면 산란 특성을 이용한 FACScan 기기로 분석할 수 있다. 유동 세포분석 검정에 추가하여 또는 그 대신에 형광 현미경을 사용한 대안적인 검정이 이용될 수 있다. 세포를 상기한 바와 같이 정확하게 염색하고 형광 현미경으로 검사할 수 있다. 상기 방법은 개개의 세포의 가시화를 허용하지만, 항원의 밀도에 따라 감도를 감소시킬 수 있다.
본 발명의 LRP6 항체는 웨스턴 블로팅에 의해 LRP6 폴리펩티드 또는 항원 단편과의 반응성에 대해 추가로 시험될 수 있다. 간략하게 설명하면, 정제된 LRP6 폴리펩티드 또는 융합 단백질, 또는 LRP6을 발현하는 세포로부터의 세포 추출물을 제조하여 나트륨 도데실 술페이트 폴리아크릴아미드 겔 전기영동을 실시할 수 있다. 전기영동 후에, 분리된 항원을 니트로셀룰로스 막으로 옮기고, 10% 소 태아 혈청으로 차단하고, 시험할 모노클로날 항체로 프로빙한다. 인간 IgG 결합은 항-인간 IgG 알칼리성 포스파타제를 사용하여 검출할 수 있고, BCIP/NBT 기질 정제 (시그마 케미칼 컴퍼니 (Sigma Chem. Co.), 미국 미주리주 세인트 루이스)로 발색시킬 수 있다. 기능성 검정의 예는 아래 실시예 섹션에 또한 설명되어 있다.
예방 및
치료적
용도
본 발명은 LRP6 Wnt 신호 전달 경로와 연관된 질환 또는 장애의 치료를 필요로 하는 대상체에게 유효량의 본 발명의 항체를 투여함으로써 LRP6 Wnt 신호 전달 경로와 연관된 질환 또는 장애의 치료 방법을 제공한다. 특정 실시양태에서, 본 발명은 암의 치료 또는 예방을 필요로 하는 대상체에게 유효량의 본 발명의 항체를 투여함으로써 암 (예를 들어, 유방암, 폐암, 다발성 골수종, 난소암, 간암, 방광암, 위암, 전립선암, 급성 골수성 백혈병, 만성 골수성 백혈병, 골육종, 편평세포 암종, 및 흑색종을 치료 또는 예방하는 방법을 제공한다. 몇몇 실시양태에서, 본 발명은 Wnt 신호 전달 경로와 연관된 암의 치료 또는 예방을 필요로 하는 대상체에게 유효량의 본 발명의 항체를 투여함으로써 Wnt 신호 전달 경로와 연관된 암을 치료 또는 예방하는 방법을 제공한다.
한 실시양태에서, 본 발명은 유방암, 폐암, 다발성 골수종, 난소암, 방광암, 간암, 위암, 전립선암, 급성 골수성 백혈병, 만성 골수성 백혈병, 골육종, 편평세포 암종, 및 흑색종을 포함하고 이로 제한되지 않는, Wnt 신호 전달 경로와 연관된 암의 치료 방법을 제공한다.
LRP6 항체는 또한 골다공증, 골관절염, 다낭성 신장 질환, 당뇨병, 정신분열증, 혈관 질환, 심장 질환, 비-발암성 증식성 질환, 섬유증, 및 신경변성 질환, 예컨대 알츠하이머 질환을 포함하고 이로 제한되지 않는, 비정상적인 또는 결함이 있는 Wnt 신호 전달과 연관된 다른 장애의 치료 또는 예방을 위해 사용될 수 있다. Wnt 신호 전달 경로는 조직 복구 및 재생에서 중요한 역할을 수행한다. Wnt 신호 전달에 대해 세포를 감작화하는 작용제는 특히 많은 병태, 예컨대 골 질환, 점막염, 급성 및 만성 신장 손상에서 조직 재생을 촉진하기 위해 사용될 수 있다.
LRP6 항체를 사용한 조합 치료에 적합한 작용제는 정형적 Wnt 신호 전달 경로의 활성을 조정할 수 있는 당업계에 공지된 표준 치료제 (예를 들어, PI3 키나제 작용제)를 포함한다.
진단 용도
한 측면에서, 본 발명은 생물학적 샘플 (예를 들어, 혈액, 혈청, 세포, 조직)에서 또는 암으로 고통받거나 암 발생의 위험이 있는 개체로부터 LRP6 단백질 및/또는 핵산 발현 및 LRP6 단백질 기능을 결정하기 위한 진단 검정을 포함한다.
진단 검정, 예컨대 경쟁 검정은 공통 결합 파트너 상의 제한된 수의 결합 부위에 대해 시험 샘플 분석물과 경쟁하는 표지된 유사체 ("추적자 (tracer)")의 능력에 의지한다. 결합 파트너는 일반적으로 경쟁 전 또는 후에 불용성이고, 이어서 결합 파트너에 결합된 추적자 및 분석물은 비결합된 추적자 및 분석물로부터 분리된다. 상기 분리는 경사분리 (결합 파트너가 사전에 불용화된 경우) 또는 원심분리 (결합 파트너가 경쟁 반응 후에 침전된 경우)에 의해 달성된다. 시험 샘플 분석물의 양은 마커 물질의 양으로 결정되는 바와 같은 결합된 추적자의 양에 반비례한다. 공지된 양의 분석물을 이용한 용량-반응 곡선을 작성하고, 이것을 시험 결과와 비교하여 시험 샘플에 존재하는 분석물의 양을 정량적으로 결정한다. 이러한 검정은, 검출가능 마커로서 효소가 사용되는 경우에 ELISA 시스템이라 지칭된다. 이러한 형태의 검정에서, 항체와 LRP6 항체 사이의 경쟁적 결합으로 인해 결합된 LRP6 단백질, 바람직하게는 본 발명의 LRP6 에피토프가 생성되는데, 이는 혈청 샘플 중의 항체, 가장 특히 혈청 샘플 중의 억제 항체의 척도가 된다.
상기 검정의 유의한 이점은 억제 항체 (즉, LRP6 단백질, 구체적으로는 에피토프의 결합을 방해하는 항체)를 직접 측정한다는 점이다. 특히 ELISA 시험 형태의 이러한 검정은 임상 환경 및 통상적인 혈액 스크리닝에 상당한 정도로 적용되고 있다.
본 발명은 또한 개체가 보체 경로 활성의 조절 이상과 연관된 장애가 발생할 위험에 있는지의 여부를 결정하기 위한 예후 (또는 예측) 검정을 제공한다. 예를 들어, LRP6 유전자 내의 돌연변이를 생물학적 샘플에서 검정할 수 있다. 이러한 검정을 예후 또는 예측 목적으로 사용함으로써, LRP6 단백질, 핵산 발현 또는 활성을 특징으로 하거나 이와 연관된 장애의 발병 전에 개체를 예방적으로 처치할 수 있다.
본 발명의 또 다른 측면은 개체에서 LRP6 핵산 발현 또는 LRP6 단백질 활성을 결정함으로써 상기 개체에게 적절한 치료제 또는 예방제를 선택하는 방법을 제공한다 (본원에서 "약리유전학"이라 지칭됨). 약리유전학을 통해, 개체의 유전자형 (예를 들어, 개체가 특정 작용제에 대해 반응하는 능력을 결정하기 위해 조사한 개체의 유전자형)을 기초로 하여 개체를 치료적 또는 예방적으로 처치하기 위한 작용제 (예를 들어, 약물)를 선택할 수 한다.
본 발명의 또 다른 측면은 임상 시험에서 LRP6 단백질의 발현 또는 활성에 대한 작용제 (예를 들어, 약물)의 영향을 모니터링하는 것에 관한 것이다.
제약 조성물
LRP6 항체 (무손상 또는 결합 단편)을 포함하는 제약 또는 멸균 조성물을 제조하기 위해서, LRP6 항체 (무손상 또는 결합 단편)을 제약상 허용되는 담체 또는 부형제와 혼합한다. 조성물은 암 (유방암, 폐암, 다발성 골수종, 난소암, 간암, 방광암, 위암, 전립선암, 급성 골수성 백혈병, 만성 골수성 백혈병, 골육종, 편평세포 암종, 및 흑색종)의 치료 또는 예방에 적합한 하나 이상의 다른 치료제를 추가로 함유할 수 있다.
치료 및 진단제의 제형은 예를 들어 동결건조된 분말, 슬러리, 수용액, 로션, 또는 현탁액의 형태로 생리학상 허용되는 담체, 부형제, 또는 안정화제와 혼합함으로써 제조될 수 있다 (예를 들어, 문헌 [Hardman, et al. (2001) Goodman and Gilman's The Pharmacological Basis of Therapeutics, McGraw-Hill, New York, N.Y.]; [Gennaro (2000) Remington: The Science and Practice of Pharmacy, Lippincott, Williams, and Wilkins, New York, N.Y.]; [Avis, et al. (eds.) (1993) Pharmaceutical Dosage Forms: Parenteral Medications, Marcel Dekker, NY]; [Lieberman, et al. (eds.) (1990) Pharmaceutical Dosage Forms: Tablets, Marcel Dekker, NY]; [Lieberman, et al. (eds.) (1990) Pharmaceutical Dosage Forms: Disperse Systems, Marcel Dekker, NY]; [Weiner and Kotkoskie (2000) Excipient Toxicity and Safety, Marcel Dekker, Inc., New York, N.Y.] 참조).
치료제에 대한 투여 요법의 선택은 엔티티의 혈청 또는 조직 교체율, 증상의 수준, 엔티티의 면역원성, 및 생물학적 매트릭스에서 표적 세포의 접근성을 비롯한 몇몇의 인자에 따라 결정된다. 특정 실시양태에서, 투여 요법은 허용되는 부작용 수준에 부합하도록 환자에 전달되는 치료제의 양을 최대화한다. 따라서, 전달되는 생물학적 물질의 양은 부분적으로는 특정 엔티티 및 치료되는 병태의 중증도에 따라 결정된다. 항체, 시토카인, 및 소분자의 적절한 용량의 선택에 대한 지침이 이용가능하다 (예를 들어, 문헌 [Wawrzynczak (1996) Antibody Therapy, Bios Scientific Pub. Ltd, Oxfordshire, UK]; [Kresina (ed.) (1991) Monoclonal Antibodies, Cytokines and Arthritis, Marcel Dekker, New York, N.Y.]; [Bach (ed.) (1993) Monoclonal Antibodies and Peptide Therapy in Autoimmune Diseases, Marcel Dekker, New York, N.Y.]; [Baert, et al. (2003) New Engl. J. Med. 348:601-608]; [Milgrom, et al. (1999) New Engl. J. Med. 341:1966-1973]; [Slamon, et al. (2001) New Engl. J. Med. 344:783-792]; [Beniaminovitz, et al. (2000) New Engl. J. Med. 342:613-619]; [Ghosh, et al. (2003) New Engl. J. Med. 348:24-32]; [Lipsky, et al. (2000) New Engl. J. Med. 343:1594-1602] 참조).
적절한 용량은 예를 들어 치료에 영향을 주는 것으로 당업계에 알려지거나 의심되거나 치료에 영향을 주는 것으로 예상되는 파라미터 또는 인자를 사용하여 임상의에 의해 결정된다. 일반적으로, 용량은 최적 용량보다 다소 적은 양으로 시작하고, 목적하는 또는 최적의 효과가 임의의 부정적인 부작용에 비해 달성될 때까지 이후에 조금씩 증가시킨다. 중요한 진단 척도는 예를 들어 염증의 증상 또는 생성되는 염증성 시토카인의 수준을 포함한다.
본 발명의 제약 조성물 내의 활성 성분의 실제 투여량 수준은 환자에게 독성을 주지 않으면서 특정 환자, 조성물, 및 투여 방식에 대해 목적하는 치료 반응을 달성하기 위해 효과적인 활성 성분의 양을 얻기 위해 상이할 수 있다. 선택된 투여량 수준은 사용되는 본 발명의 특정 조성물, 또는 그의 에스테르, 염 또는 아미드의 활성, 투여 경로, 투여 시간, 사용되는 특정 화합물의 배출 속도, 치료 기간, 사용되는 특정 조성물과 조합 사용되는 다른 약물, 화합물 및/또는 물질, 치료받는 환자의 연령, 성별, 체중, 상태, 전반적인 건강 및 이전의 의료 병력 및 의료 분야에 공지된 기타 인자를 비롯한 다양한 약동학적 인자에 따라 결정될 것이다.
본 발명의 항체 또는 그의 단편을 포함하는 조성물은 연속 주입에 의해, 또는 예를 들어 1일, 1주, 또는 매주 1-7회 간격의 투여에 의해 제공될 수 있다. 용량은 정맥내, 피하, 국소, 경구, 비내, 직장, 근육내, 뇌내, 또는 흡입에 의해 제공될 수 있다. 특정 용량 프로토콜은 유의한 바람직하지 않은 부작용을 방지하는 최대 용량 또는 투여 빈도를 수반하는 것이다. 총 1주 용량은 적어도 0.05 ㎍/kg (체중), 적어도 0.2 ㎍/kg, 적어도 0.5 ㎍/kg, 적어도 1 ㎍/kg, 적어도 10 ㎍/kg, 적어도 100 ㎍/kg, 적어도 0.2 mg/kg, 적어도 1.0 mg/kg, 적어도 2.0 mg/kg, 적어도 10 mg/kg, 적어도 25 mg/kg, 또는 적어도 50 mg/kg일 수 있다 (예를 들어, 문헌 [Yang, et al. (2003) New Engl. J. Med. 349:427-434]; [Herold, et al. (2002) New Engl. J. Med. 346:1692-1698]; [Liu, et al. (1999) J. Neurol. Neurosurg. Psych. 67:451-456]; [Portielji, et al. (2003) Cancer Immunol. Immunother. 52:133-144] 참조). 항체 또는 그의 단편의 목적하는 용량은 몰/kg 체중 기준으로 항체 또는 폴리펩티드에 대한 것과 거의 동일하다. 항체 또는 그의 단편의 목적하는 혈장 농도는 대략 몰/kg 체중 기준으로 한다. 용량은 적어도 15 ㎍, 적어도 20 ㎍, 적어도 25 ㎍, 적어도 30 ㎍, 적어도 35 ㎍, 적어도 40 ㎍, 적어도 45 ㎍, 적어도 50 ㎍, 적어도 55 ㎍, 적어도 60 ㎍, 적어도 65 ㎍, 적어도 70 ㎍, 적어도 75 ㎍, 적어도 80 ㎍, 적어도 85 ㎍, 적어도 90 ㎍, 적어도 95 ㎍, 또는 적어도 100 ㎍일 수 있다. 대상체에게 투여되는 총 용량은 적어도 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 또는 12회분, 또는 그 초과일 수 있다.
본 발명의 항체 또는 그의 단편에 대해, 환자에게 투여되는 투여량은 0.0001 mg/kg 내지 100 mg/kg (환자의 체중)일 수 있다. 투여량은 0.0001 mg/kg 내지 20 mg/kg, 0.0001 mg/kg 내지 10 mg/kg, 0.0001 mg/kg 내지 5 mg/kg, 0.0001 내지 2 mg/kg, 0.0001 내지 1 mg/kg, 0.0001 mg/kg 내지 0.75 mg/kg, 0.0001 mg/kg 내지 0.5 mg/kg, 0.0001 mg/kg 내지 0.25 mg/kg, 0.0001 내지 0.15 mg/kg, 0.0001 내지 0.10 mg/kg, 0.001 내지 0.5 mg/kg, 0.01 내지 0.25 mg/kg, 또는 0.01 내지 0.10 mg/kg (환자의 체중)일 수 있다.
본 발명의 항체 또는 그의 단편의 투여량은 환자의 체중 (킬로그램(kg) 단위)에 투여되는 용량 (mg/kg 단위)을 곱함으로써 계산될 수 있다. 본 발명의 항체 또는 그의 단편의 투여량은 150 ㎍/kg 이하, 125 ㎍/kg 이하, 100 ㎍/kg 이하, 95 ㎍/kg 이하, 90 ㎍/kg 이하, 85 ㎍/kg 이하, 80 ㎍/kg 이하, 75 ㎍/kg 이하, 70 ㎍/kg 이하, 65 ㎍/kg 이하, 60 ㎍/kg 이하, 55 ㎍/kg 이하, 50 ㎍/kg 이하, 45 ㎍/kg 이하, 40 ㎍/kg 이하, 35 ㎍/kg 이하, 30 ㎍/kg 이하, 25 ㎍/kg 이하, 20 ㎍/kg 이하, 15 ㎍/kg 이하, 10 ㎍/kg 이하, 5 ㎍/kg 이하, 2.5 ㎍/kg 이하, 2 ㎍/kg 이하, 1.5 ㎍/kg 이하, 1 ㎍/kg 이하, 0.5 ㎍/kg 이하, 또는 0.5 ㎍/kg (환자의 체중) 이하일 수 있다.
본 발명의 항체 또는 그의 단편의 단위 용량은 0.1 mg 내지 20 mg, 0.1 mg 내지 15 mg, 0.1 mg 내지 12 mg, 0.1 mg 내지 10 mg, 0.1 mg 내지 8 mg, 0.1 mg 내지 7 mg, 0.1 mg 내지 5 mg, 0.1 내지 2.5 mg, 0.25 mg 내지 20 mg, 0.25 내지 15 mg, 0.25 내지 12 mg, 0.25 내지 10 mg, 0.25 내지 8 mg, 0.25 mg 내지 7 mg, 0.25 mg 내지 5 mg, 0.5 mg 내지 2.5 mg, 1 mg 내지 20 mg, 1 mg 내지 15 mg, 1 mg 내지 12 mg, 1 mg 내지 10 mg, 1 mg 내지 8 mg, 1 mg 내지 7 mg, 1 mg 내지 5 mg, 또는 1 mg 내지 2.5 mg일 수 있다.
본 발명의 항체 또는 그의 단편의 투여량은 대상체에서 적어도 0.1 ㎍/ml, 적어도 0.5 ㎍/ml, 적어도 1 ㎍/ml, 적어도 2 ㎍/ml, 적어도 5 ㎍/ml, 적어도 6 ㎍/ml, 적어도 10 ㎍/ml, 적어도 15 ㎍/ml, 적어도 20 ㎍/ml, 적어도 25 ㎍/ml, 적어도 50 ㎍/ml, 적어도 100 ㎍/ml, 적어도 125 ㎍/ml, 적어도 150 ㎍/ml, 적어도 175 ㎍/ml, 적어도 200 ㎍/ml, 적어도 225 ㎍/ml, 적어도 250 ㎍/ml, 적어도 275 ㎍/ml, 적어도 300 ㎍/ml, 적어도 325 ㎍/ml, 적어도 350 ㎍/ml, 적어도 375 ㎍/ml, 또는 적어도 400 ㎍/ml의 혈청 역가를 달성할 수 있다. 별법으로, 본 발명의 항체 또는 그의 단편의 투여량은 대상체에서 적어도 0.1 ㎍/ml, 적어도 0.5 ㎍/ml, 적어도 1 ㎍/ml, 적어도, 2 ㎍/ml, 적어도 5 ㎍/ml, 적어도 6 ㎍/ml, 적어도 10 ㎍/ml, 적어도 15 ㎍/ml, 적어도 20 ㎍/ml, 적어도 25 ㎍/ml, 적어도 50 ㎍/ml, 적어도 100 ㎍/ml, 적어도 125 ㎍/ml, 적어도 150 ㎍/ml, 적어도 175 ㎍/ml, 적어도 200 ㎍/ml, 적어도 225 ㎍/ml, 적어도 250 ㎍/ml, 적어도 275 ㎍/ml, 적어도 300 ㎍/ml, 적어도 325 ㎍/ml, 적어도 350 ㎍/ml, 적어도 375 ㎍/ml, 또는 적어도 400 ㎍/ml의 혈청 역가를 달성할 수 있다.
본 발명의 항체 또는 그의 단편의 용량은 반복 투여될 수 있고, 투여는 적어도 1일, 2일, 3일, 5일, 10일, 15일, 30일, 45일, 2개월, 75일, 3개월 또는 적어도 6개월 간격으로 시행될 수 있다.
특정 환자에 대한 유효량은 치료되는 병태, 환자의 전반적인 건강, 방법 경로 및 투여 용량 및 부작용의 정도와 같은 요인에 따라 상이할 수 있다 (예를 들어, 문헌 [Maynard, et al. (1996) A Handbook of SOPs for Good Clinical Practice, Interpharm Press, Boca Raton, Fla.]; [Dent (2001) Good Laboratory and Good Clinical Practice, Urch Publ., London, UK] 참조).
투여 경로는 예를 들어 피부 적용, 정맥내, 복강내, 뇌내, 근육내, 안내, 동맥내, 뇌척수내, 병변내 주사 또는 주입, 또는 지속 방출 시스템 또는 임플란트에 의한 것일 수 있다 (예를 들어, 문헌 [Sidman et al. (1983) Biopolymers 22:547-556]; [Langer, et al. (1981) J. Biomed. Mater. Res. 15:167-277]; [Langer (1982) Chem. Tech. 12:98-105]; [Epstein, et al. (1985) Proc. Natl. Acad. Sci. USA 82:3688-3692]; [Hwang, et al. (1980) Proc. Natl. Acad. Sci. USA 77:4030-4034]; 미국 특허 번호 6,350,466 및 6,316,024 참조). 필요한 경우에, 조성물은 또한 주사 부위의 통증을 완화하기 위해 가용화제 및 국소 마취제, 예컨대 리도카인을 포함할 수 있다. 또한, 예를 들어 흡입기 또는 연무기 (nebulizer)의 사용, 및 에어로졸화제를 사용한 제형화에 의한 폐내 투여도 사용될 수 있다. 예를 들어, 각각 그 전체가 본원에 참조로 포함되는 미국 특허 번호 6,019,968, 5,985,320, 5,985,309, 5,934,272, 5,874,064, 5,855,913, 5,290,540, 및 4,880,078; 및 PCT 공개 WO 92/19244, WO 97/32572, WO 97/44013, WO 98/31346 및 WO 99/66903을 참조한다.
또한, 본 발명의 조성물은 당업계에 공지된 하나 이상의 다양한 방법을 사용하여 하나 이상의 투여 경로를 통해 투여될 수 있다. 당업자에게 이해되는 바와 같이, 투여 경로 및/또는 방식은 목적하는 결과에 따라 상이할 것이다. 본 발명의 항체 또는 그의 단편에 대해 선택된 투여 경로는 예를 들어 주사 또는 주입에 의한 정맥내, 근육내, 피부내, 복강내, 피하, 척수 또는 다른 비경구 투여 경로를 포함한다. 비경구 투여는 장내 및 국소 투여 이외의 다른, 대체로 주사에 의한 투여 방식을 나타내고, 정맥내, 근육내, 동맥내, 경막내, 낭내, 안와내, 심장내, 피부내, 복강내, 경기관, 피하, 표피하, 관절내, 낭하, 거미막하, 척수내, 경막외 및 흉골내 주사 및 주입을 포함하고 이로 제한되지 않는다. 별법으로, 본 발명의 조성물은 비-비경구 경로를 통해, 예컨대 국소, 표피 또는 점막 투여 경로, 예를 들어, 비내, 경구, 질, 직장, 설하 또는 국소 투여될 수 있다. 한 실시양태에서, 본 발명의 항체 또는 그의 단편은 주입에 의해 투여된다. 또 다른 실시양태에서, 본 발명의 다중특이적 에피토프 결합 단백질은 피하 투여된다.
본 발명의 항체 또는 그의 단편이 제어 방출 또는 지속 방출 시스템으로 투여될 경우, 제어 또는 지속 방출을 달성하기 위해 펌프가 사용될 수 있다 ([Langer, 상기 문헌]; [Sefton, 1987, CRC Crit. Ref Biomed. Eng. 14:20]; [Buchwald et al., 1980, Surgery 88:507]; [Saudek et al., 1989, N. Engl. J. Med. 321:574] 참조). 중합체 물질이 본 발명의 치료제의 제어 또는 지속 방출을 달성하기 위해 사용될 수 있다 (예를 들어, 문헌 [Medical Applications of Controlled Release, Langer and Wise (eds.), CRC Pres., Boca Raton, Fla. (1974)]; [Controlled Drug Bioavailability, Drug Product Design and Performance, Smolen and Ball (eds.), Wiley, New York (1984)]; [Ranger and Peppas, 1983, J., Macromol. Sci. Rev. Macromol. Chem. 23:61]; 또한 문헌 [Levy et al., 1985, Science 228:190]; [During et al., 1989, Ann. Neurol. 25:351]; [Howard et al., 1989, J. Neurosurg. 71:105]); 미국 특허 번호 5,679,377; 미국 특허 번호 5,916,597; 미국 특허 번호 5,912,015; 미국 특허 번호 5,989,463; 미국 특허 번호 5,128,326; PCT 공개 WO 99/15154; 및 PCT 공개 WO 99/20253 참조). 지속 방출 제형에 사용되는 중합체의 예는 폴리(2-히드록시 에틸 메타크릴레이트), 폴리(메틸 메타크릴레이트), 폴리(아크릴산), 폴리(에틸렌-코-비닐 아세테이트), 폴리(메타크릴산), 폴리글리콜라이드 (PLG), 폴리무수물 (polyanhydride), 폴리(N-비닐 피롤리돈), 폴리(비닐 알콜), 폴리아크릴아미드, 폴리(에틸렌 글리콜), 폴리락티드 (PLA), 폴리(락티드-co-글리콜리드) (PLGA), 및 폴리오르토에스테르를 포함하고 이로 제한되지 않는다. 한 실시양태에서, 지속 방출 제형에 사용되는 중합체는 불활성이고, 여과할 수 있는 불순물이 존재하지 않고, 저장시 안정하고, 멸균 상태이고, 생분해성이다. 제어 또는 지속 방출 시스템은 예방 또는 치료 표적 근처에 위치할 수 있고, 따라서 전신 용량의 일부만을 필요로 한다 (예를 들어, 문헌 [Goodson, in Medical Applications of Controlled Release, 상기 문헌, vol. 2, pp. 115-138 (1984)] 참조).
제어 방출 시스템은 문헌 [Langer, 1990, Science 249:1527-1533]에서 논의된 바 있다. 당업자에게 공지된 임의의 기술을 사용하여 하나 이상의 본 발명의 항체 또는 그의 단편을 포함하는 지속 방출 제형을 생산할 수 있다. 예를 들어, 각각 그 전체가 본원에 참조로 포함되는 미국 특허 번호 4,526,938, PCT 공개 WO 91/05548, PCT 공개 WO 96/20698, 문헌 [Ning et al., 1996, "Intratumoral Radioimmunotheraphy of a Human Colon Cancer Xenograft Using a Sustained-Release Gel," Radiotherapy & Oncology 39:179-189], [Song et al., 1995, "Antibody Mediated Lung Targeting of Long-Circulating Emulsions," PDA Journal of Pharmaceutical Science & Technology 50:372-397], [Cleek et al., 1997, "Biodegradable Polymeric Carriers for a bFGF Antibody for Cardiovascular Application," Pro. Int'l. Symp. Control. Rel. Bioact. Mater. 24:853-854], 및 [Lam et al., 1997, "Microencapsulation of Recombinant Humanized Monoclonal Antibody for Local Delivery," Proc. Int'l. Symp. Control Rel. Bioact. Mater. 24:759-760]을 참조한다.
본 발명의 항체 또는 그의 단편이 국소 투여될 경우, 이것은 연고, 크림, 경피 패치, 로션, 겔, 샴푸, 스프레이, 에어로졸, 용액, 에멀젼, 또는 당업자에게 공지된 다른 형태로 제형화될 수 있다. 예를 들어, 문헌 [Remington's Pharmaceutical Sciences and Introduction to Pharmaceutical Dosage Forms, 19th ed., Mack Pub. Co., Easton, Pa. (1995)]을 참조한다. 비-분무가능 국소 투여 형태의 경우, 국소 적용에 적합한 담체 또는 하나 이상의 부형제를 포함하고 몇몇 경우에 물보다 큰 동적 점도를 갖는 점성 내지 반고체 또는 고체 형태가 일반적으로 사용된다. 적합한 제형은, 필요한 경우 멸균되거나 예를 들어 삼투압과 같은 다양한 특성에 영향을 주는 보조제 (예를 들어, 보존제, 안정화, 습윤제, 완충제, 또는 염)와 혼합되는 용액, 현탁액, 에멀젼, 크림, 연고, 분말, 도찰제 (liniment), 고약 등을 포함하고 이로 제한되지 않는다. 다른 적합한 국소 투여 형태는 몇몇 경우에 활성 성분이 고체 또는 액체 불활성 담체와 조합되어 압축 휘발물질 (예를 들어, 기상 추진제, 예컨대 프레온)의 혼합물에 또는 눌러 짜내는 플라스틱 용기 (squeeze bottle)에 충전되는 분무가능 에어로졸 제제를 포함한다. 보습제 또는 습윤제가 또한 필요한 경우 제약 조성물 및 투여 형태에 첨가될 수 있다. 상기 추가의 성분의 예는 당업계에 공지되어 있다.
항체 또는 그의 단편을 포함하는 조성물이 비내 투여될 경우, 이것은 에어로졸 형태, 스프레이, 연무제 (mist) 또는 점적제의 형태로 제형화될 수 있다.
제2 치료제, 예를 들어 시토카인, 스테로이드, 화학치료제, 항생제, 또는 방사선과 함께 동시 투여하거나 치료하기 위한 방법은 당업계에 공지되어 있다 (예를 들어, 문헌 [Hardman, et al. (eds.) (2001) Goodman and Gilman's The Pharmacological Basis of Therapeutics, 10th ed., McGraw-Hill, New York, N.Y.]; [Poole and Peterson (eds.) (2001) Pharmacotherapeutics for Advanced Practice: A Practical Approach, Lippincott, Williams & Wilkins, Phila., Pa.]; [Chabner and Longo (eds.) (2001) Cancer Chemotheraphy 및 Biotheraphy, Lippincott, Williams & Wilkins, Phila., Pa.] 참조). 유효량의 치료제는 증상을 적어도 10%; 적어도 20%; 적어도 약 30%; 적어도 40%, 또는 적어도 50% 감소시킬 수 있다.
본 발명의 항체 또는 그의 단편과 조합하여 투여될 수 있는 추가의 요법제 (예를 들어, 예방제 또는 치료제)는 본 발명의 항체 또는 그의 단편과 5분 미만, 30분 미만, 1시간, 약 1시간, 약 1 내지 약 2시간, 약 2시간 내지 약 3시간, 약 3시간 내지 약 4시간, 약 4시간 내지 약 5시간, 약 5시간 내지 약 6시간, 약 6시간 내지 약 7시간, 약 7시간 내지 약 8시간, 약 8시간 내지 약 9시간, 약 9시간 내지 약 10시간, 약 10시간 내지 약 11시간, 약 11시간 내지 약 12시간, 약 12시간 내지 18시간, 18시간 내지 24시간, 24시간 내지 36시간, 36시간 내지 48시간, 48시간 내지 52시간, 52시간 내지 60시간, 60시간 내지 72시간, 72시간 내지 84시간, 84시간 내지 96시간, 또는 96시간 내지 120시간의 간격으로 투여될 수 있다. 2개 이상의 요법제가 1회의 동일 환자 방문시에 투여될 수 있다.
본 발명의 항체 또는 그의 단편 및 다른 요법제는 주기적으로 투여될 수 있다. 주기적 요법은 일정 기간 동안의 제1 요법제 (예를 들어, 제1 예방제 또는 치료제)의 투여, 이어서 일정 기간 동안의 제2 요법제 (예를 들어, 제2 예방제 또는 치료제)의 투여, 이어서 일정 기간 동안의 제3 요법제 (예를 들어, 제3 예방제 또는 치료제)의 투여 등, 및 상기 순차적인 투여의 반복, 즉, 요법제 중 하나에 대한 내성의 발생을 감소시키고, 요법제 중 하나의 부작용을 방지 또는 감소시키고/시키거나, 요법제의 효능을 개선하기 위한 순환을 포함한다.
특정 실시양태에서, 본 발명의 항체 또는 그의 단편은 생체 내에서 적합한 분포를 보장하도록 제형화될 수 있다. 예를 들어, 혈액-뇌 장벽 (BBB)은 많은 고친수성 화합물을 차단한다. 본 발명의 치료 화합물이 BBB를 가로지르는 것을 보장하기 위해 (원하는 경우에), 이들은 예를 들어 리포솜으로 제형화될 수 있다. 리포솜 제조 방법에 대해서는, 예를 들어, 미국 특허 번호 4,522,811; 5,374,548; 및 5,399,331을 참조한다. 리포솜은 특이적 세포 또는 장기 내로 선택적으로 수송되는 하나 이상의 모이어티를 포함하고, 따라서 표적화 약물 전달을 향상시킬 수 있다 (예를 들어, 문헌 [V. V. Ranade (1989) J. Clin. Pharmacol. 29:685] 참조). 예시적인 표적화 모이어티는 엽산 또는 비오틴 (예를 들어, 미국 특허 번호 5,416,016 (Low et al.)); 만노시드 (Umezawa et al., (1988) Biochem. Biophys. Res. Commun. 153:1038); 항체 ([P. G. Bloeman et al. (1995) FEBS Lett. 357:140]; [M. Owais et al. (1995) Antimicrob. Agents Chemother. 39:180]); 계면활성제 단백질 A 수용체 (Briscoe et al. (1995) Am. J. Physiol. 1233:134); p120 (Schreier et al (1994) J. Biol. Chem. 269:9090); 또한, 문헌 [K. Keinanen, M. L. Laukkanen (1994) FEBS Lett. 346:123]; [J. J. Killion; I.J. Fidler (1994) Immunomethods 4:273] 참조)을 포함한다.
본 발명은 본 발명의 항체 또는 그의 단편을 포함하는 제약 조성물을 단독으로 또는 다른 요법제와 조합하여 그를 필요로 하는 대상체에게 투여하기 위한 프로토콜을 제공한다. 본 발명의 조합 요법의 요법제 (예를 들어, 예방제 또는 치료제)는 대상체에게 동시에 또는 순차적으로 투여될 수 있다. 본 발명의 조합 요법의 요법제 (예를 들어, 예방제 또는 치료제)는 또한 주기적으로 투여될 수 있다. 주기적 요법은 일정 기간 동안의 제1 요법제 (예를 들어, 제1 예방제 또는 치료제)의 투여, 이어서 일정 기간 동안의 제2 요법제 (예를 들어, 제2 예방제 또는 치료제)의 투여 및 상기 순차적인 투여의 반복, 즉, 요법제 (예를 들어, 작용제) 중 하나에 대한 내성의 발생을 감소시키고, 요법제 (예를 들어, 작용제) 중 하나의 부작용을 방지 또는 감소시키고/시키거나, 요법제의 효능을 개선하기 위한 순환을 포함한다.
본 발명의 조합 요법의 요법제 (예를 들어, 예방제 또는 치료제)는 대상체에게 동시에 투여할 수 있다. 용어 "동시에"는 정확히 동일한 시간에 요법제 (예를 들어, 예방 또는 치료제)의 투여로 제한되지 않고, 본 발명의 항체 또는 그의 단편이 달리 투여된 경우보다 증가된 이익을 제공하기 위해 다른 요법제(들)과 함께 작용할 수 있는 순서 및 시간 간격으로 본 발명의 항체 또는 그의 단편을 포함하는 제약 조성물이 대상체에게 투여됨을 의미한다. 예를 들어, 각각의 요법제는 동시에 또는 상이한 시점에서 임의의 순서로 순차적으로 대상체에게 투여될 수 있지만; 동시에 투여되지 않을 경우에도, 이들은 목적하는 치료 또는 예방 효과를 제공하기 위해 충분히 가까운 시간 간격으로 투여되어야 한다. 각각의 요법제는 임의의 적절한 형태로 및 임의의 적합한 경로에 의해 대상체에게 별개로 투여될 수 있다. 다양한 실시양태에서, 요법제 (예를 들어, 예방제 또는 치료제)는 대상체에게 15분 미만, 30분 미만, 1시간 미만, 약 1시간, 약 1 내지 약 2시간, 약 2시간 내지 약 3시간, 약 3시간 내지 약 4시간, 약 4시간 내지 약 5시간, 약 5시간 내지 약 6시간, 약 6시간 내지 약 7시간, 약 7시간 내지 약 8시간, 약 8시간 내지 약 9시간, 약 9시간 내지 약 10시간, 약 10시간 내지 약 11시간, 약 11시간 내지 약 12시간, 24시간, 48시간, 72시간, 또는 1주의 간격으로 투여될 수 있다. 다른 실시양태에서, 2개 이상의 요법제 (예를 들어, 예방제 또는 치료제)는 환자 방문시에 투여된다.
조합 요법의 예방제 또는 치료제는 대상체에게 동일한 제약 조성물 내에서 투여될 수 있다. 별법으로, 조합 요법의 예방제 또는 치료제는 대상체에게 별개의 제약 조성물 내에서 동시에 투여할 수 있다. 예방 또는 치료제는 대상체에게 동일한 또는 상이한 투여 경로로 투여될 수 있다.
본 발명은 충분히 설명되었지만, 예시적인 것으로서 추가로 제한하는 것을 의미하지 않는 다음 실시예 및 특허청구범위에 의해 추가로 예시된다.
실시예
방법 및 물질
1:
패닝
, 항체 확인 및 특징 결정
(a)
패닝
(i)
HuCAL
GOLD
®
패닝
인간 LRP6을 인식하는 항체의 선택을 위해, 몇몇 패닝 전략을 적용하였다. 인간 LRP6 단백질에 대한 치료적 항체는 항체 변이체 단백질의 공급원으로서 상업상 이용가능한 파지 디스플레이 라이브러리인 MorphoSys HuCAL GOLD® 라이브러리를 사용하여 높은 결합 친화도를 가진 클론의 선택에 의해 생성하였다.
파지미드 라이브러리는 HuCAL® 개념 (Knappik et al., (2000) J Mol Biol 296:57-86)에 기반하고, Fab를 파지 표면 상에 디스플레이하기 위해 시스디스플레이(CysDisplay)™ 기술을 이용한다 (WO01/05950 (로닝(Lohning))).
상세히 설명하면, 현재 계획에서 사용된 HuCAL GOLD® 라이브러리는 문헌 [Rothe et al., (2007) J Mol Biol]에 설명되어 있다. 항-LRP6 항체의 선택을 위해 헬퍼 파지 VCSM13를 사용하여 생산된 파지미드 또는 하이퍼파지를 사용하여 생산된 파지미드 (Rondot et al., (2001) Nat Biotechnol 19:75-78)를 사용하였다.
(
ii
)
LRP6
에 대한 전체 세포
패닝
전체 세포 패닝을 위해, eGFP에 융합된 LRP6의 아미노 말단 단편 (아미노산 1-1482)을 발현하는 세포주 HEK293-hLRP6ΔC-eGFP를 사용하였다. 특이적 HuCAL GOLD® 항체 파지미드를 LRP6 발현 세포주로의 인큐베이션 후에 용리시킨 다음, HEK293-LRP5/6-shRNA 세포에 사후-흡착시켰다. 생성되는 HuCAL 파지-함유 상청액을 이. 콜라이 TG1 세포 상에서 적정하고, 헬퍼 파지를 사용한 이. 콜라이 TG1 세포의 감염 후에 구조하였다. 폴리클로날 증폭된 파지 출력 (output)을 다시 적정하고, 연속적인 선택 단계에서 사용하였다.
(
iii
)
LRP6
에 대한
Fc
포획
패닝
Fc 포획 패닝을 위해, 차단된 파지를 Fc-포획된 LRP6-Fc와 함께 인큐베이션하고, 비특이적 파지를 상이한 시간에 상이한 농도의 PBST, 및 PBS를 사용하여 세척 제거하였다.
남아있는 파지를 용리시키고, 이. 콜라이 TG1 박테리아의 감염을 위해 즉시 사용하였다. 증폭, 파지 생산 및 출력 역가 결정을 LRP6에 대한 전체 세포 패닝에서 상기 설명된 바와 같이 수행하였다.
(
iv
)
LRP6
에 대한 차별적 전체 세포
패닝
HEK293-hLRP6ΔC-eGFP 세포 상의 항체 선택 및 재조합 인간 LRP6-Fc에 대한 선택과 함께 차별적 전체 세포 패닝을 전체 세포 패닝에 대해 및 Fc 포획 패닝에 대해 상기 설명된 바와 같이 수행하였다.
선택 과정 동안, 상이한 시간 기간 동안 상이한 농도의 PBS 및 PBST를 사용하여 비특이적 파지를 제거하였다.
이. 콜라이 TG1의 파지 감염, 증폭, 파지 생산 및 출력 역가 결정을 LRP6에 대한 전체 세포 패닝에서 상기 설명된 바와 같이 수행하였다.
(v)
LRP6
에 대한 반-용액 (
Semi
-
Solution
)
패닝
반-용액 패닝을 위해, 재조합 인간 LRP6-Fc 및 BSA를 토실 (tosyl) 활성화된 M-280-디나비드 (Dynabead)에 공유 연결시켰다.
예비-청소한 파지를 회전기 상에서 LRP6-코팅된 비드와 함께 인큐베이션하였다. 이어서, 비드를 자력 분리기를 사용하여 수집하고, PBST 및 PBS로 세척하였다. 비드-결합된 파지를 용리시키고, 이. 콜라이 TG1 박테리아의 감염을 위해 즉시 사용하였다. 파지 감염, 증폭, 파지 생산 및 출력 역가 결정을 LRP6에 대한 전체 세포 패닝에서 상기 설명된 바와 같이 수행하였다.
(b) 선택된
Fab
단편의
서브클로닝
및 미세발현
가용성 Fab의 신속한 발현을 촉진하기 위해, 선택된 HuCAL GOLD® 파지의 Fab 코딩 삽입물을 발현 벡터 pMORPH®X9_FH 또는 pMORPH®X9_FS 내로 서브클로닝하였다. TG1-F의 형질전환 후에, 단일 클론 발현, 및 HuCAL®-Fab 단편을 함유하는 주변세포질 추출물의 제조를 이전에 설명된 바와 같이 수행하였다 (Rauchenberger et al., (2003)).
2: 스크리닝
(i)
HEK293
-
hLRP6
ΔC-
eGFP
세포에 대한
FACS
스크리닝
패닝 전략인 전체 세포 패닝, 차별적 전체 세포 패닝, 및 반-용액 패닝에 의해 선택된 클론을 반대스크리닝을 위해 HEK293-hLRP6ΔC-eGFP 세포 및 HEK293-LRP5/6-shRNA 세포에 대한 유동 세포분석에 의해 스크리닝하였다. 상기 설명된 바와 같은 Fc 포획 패닝 전략의 일차 적중치를 또한 유동 세포측정법에 의해 시험할 수 있다.
세포를 70 내지 80% 융합도 (confluency)에서 수거하고, FACS 완충제 내에 재현탁시키고, 96웰 U자형 마이크로타이터 플레이트 내에서 박테리아 세포 용해물로 염색하였다. 항체 결합은 형광색소-접합된 검출 항체를 사용하여 밝혀냈다. 염색된 세포를 2회 세척하고, FACSArray 기기 (벡톤디킨슨 (BectonDickinson))를 사용하여 평균 형광 강도를 측정하고 분석하였다.
(
ii
) 재조합 인간
LRP6
-
Fc
에 대한
Fc
-포획 스크리닝
Fc-포획 패닝에서 선택된 클론을 Fc-포획-ELISA 구성에서 스크리닝하였다. 맥시소르프 (Maxisorp; 눈크 (Nunc, 미국 뉴욕주 로체스터)) 384 웰 플레이트를 염소 항-인간 IgG, Fc 단편 특이적 항체로 코팅하였다. PBST로 세척하고 웰을 차단한 후에, 재조합 인간 LRP6-Fc를 첨가하였다. 코팅된 플레이트를 세척한 후에, 세포 용해물을 첨가하고, 기질 AttoPhos와 함께 AP-접합된 염소 IgG 항-인간 IgG F(ab')2를 사용하여 결합된 Fab 단편을 검출하였다. 형광을 데칸 (Tecan) 플레이트 판독기를 사용하여 535 nm에서 판독하였다.
3: 이.
콜라이
내에서
HuCAL
®-
Fab
항체의 발현 및 정제
TG-1 세포 내에서 pMORPH®X9_Fab_FH 또는 pMORPH®X11_Fab_FH에 의해 코딩되는 Fab 단편의 발현을 IPTG의 첨가에 의해 유도하였다. 세포를 리소자임을 사용하여 파괴하고, Fab 단편을 Ni-NTA 크로마토그래피 (바이오-라드 (Bio-Rad), 독일)에 의해 단리하였다. 단백질 농도는 UV-분광광도법에 의해 결정하였다. Fab 단편의 순도는 SDS-PAGE를 이용하여 변성된, 환원된 상태에서 및 HP-SEC에 의해 천연 상태에서 분석하였다.
4: 친화도 결정
(i) 표면
플라스몬
공명 측정
KD 값의 결정을 위해, 표면 플라스몬 공명 기술을 적용하였다. LRP6-Fc-융합체를 포획하기 위해 항-인간-Fc-포획 CM5 칩 (비아코어, 스웨덴)을 사용한 후, 상이한 농도에서 리간드 (Fab)를 주사하였다.
(
ii
) 섹터
이미져
(
Sector
Imager
) 6000 (
MSD
)을 사용한
K
D
결정을 위한 용액 평형 적정 (
SET
) 방법
용액 평형 적정 (SET)에 의한 KD 결정을 위해, 항체 단백질의 단량체 분획을 사용하였다 (적어도 90% 단량체 함량, 분석적 SEC에 의해 분석함; 각각 Fab에 대해 수퍼덱스75 (Superdex75; 아머샴 파마시아 (Amersham Pharmacia)) 또는 IgG에 대해 토소 (Tosoh) G3000SWXL (토소 바이오사시언스 (Tosoh Bioscience)).
용액 내에서 친화도 결정은 기본적으로 문헌 (Friguet et al., (1985) J Immunol Methods 77:305-319)에 설명된 바와 같이 수행하였다. SET 방법의 민감도 및 정확도를 개선하기 위해, 전통적인 ELISA에서 ECL 기반 기술로 옮겨졌다 (Haenel et al., (2005) Anal Biochem. 339: 182-184).
데이터는 주문제작된 피팅 (fitting) 모델을 적용하는 XLfit (IDBS) 소프트웨어를 사용하여 평가하였다. Fab 분자의 KD 결정을 위해, 피트 모델을 사용하였다 (문헌 [Abraham et al. (1996) Journal of Molecular Recognition 9:456-461]에 따라 변형된 [Haenel et al., 상기 문헌]에 따라).
[Fab]t: 적용된 총 Fab 농도
x: 적용된 총 가용성 항원 농도 (결합 부위)
Bmax: 항원 없이 Fab의 최대 신호
KD: 친화도.
5: 친화도 성숙 후의 스크리닝
HEK293T
/17 세포 상에서
EC
50
결정
EC50 값을 FACS 측정으로 모 HEK293T/17 세포 상에서 결정하였다. 일반적인 항체 적정 곡선은 10 내지 12개의 상이한 항체 희석액을 포함하고, 적정은 약 150 내지 200 ㎍/ml (최종 농도)의 농도에서 시작하였다. 세포를 아큐타스(Accutase)를 사용하여 수거하고, FACS 완충제 내에 재현탁시키고, 96-웰 플레이트의 웰에 분포하고, 항체 희석액으로 염색하였다. EC50 값은 비-선형 회귀 분석을 사이용하여 프로그램 그래프패드 프리즘 (GraphPad Prism)을 이용하여 결정하였다.
6:
IgG
로의 전환
전장 IgG를 발현하기 위해, 중쇄 (VH) 및 경쇄 (VL)의 가변 도메인 단편은 Fab 발현 벡터로부터 인간 IgG2, 인간 IgG4, 인간 IgG4_Pro, 및 인간 IgG1fLALA에 대해 적절한 pMORPH®_hIg 벡터 내로 서브클로닝하였다.
7: 인간
IgG
의 일시적 발현 및 정제
진핵 HKB11 세포를 등량의 IgG 중쇄 및 경쇄 발현 벡터 (pMORPH2) 또는 IgG의 중쇄 및 경쇄를 코딩하는 발현 벡터 DNA (pMORPH4)로 형질감염시켰다. 멸균 여과 후에, 용액을 표준 단백질 A 친화도 크로마토그래피 (맵셀렉트 슈어(MabSelect SURE), 지이 헬쓰케어 (GE Healthcare))로 처리하였다. 단백질 농도는 UV-분광광도법에 의해 결정하였다. IgG의 순도는 변성, 환원 및 비-환원 조건 하에 SDS-PAGE에서 또는 애질런트 바이오애널라이저 (Agilent BioAnalyzer)를 사용함으로써 천연 상태에서 HP-SEC에 의해 분석하였다.
8:
Wnt
리포터 유전자 검정
Wnt 신호 전달을 억제하는 항-LRP6 항체의 능력을 Wnt1 및 Wnt3a 반응성 루시퍼라제 리포터 유전자 검정으로 시험하였다. 세포를 Wnt3a 조건화 배지로 또는 Wnt1, Wnt3a, 또는 다른 Wnt 발현 플라스미드의 동시-형질감염에 의해 자극하였다.
(i) 조건화 배지를 사용한
Wnt3a
리포터 유전자 검정
104개 HEK293-STF 세포/웰을 96웰 조직 배양 플레이트 내에 접종하고, 세포를 100 ㎕ 배지 내에서 37℃/5% CO2에서 밤새 인큐베이션하였다.
다음날, 다양한 항-LRP6 항체 희석액 및 DKK1 희석액 (양성 대조군)을 순수하게 또는 Wnt3a-조건화 배지 내에 희석시켜 제조하고, 60 ㎕/웰의 상청액을 96웰 조직 배양 플레이트로부터 제거하고, 60 ㎕/웰의 조건화 배지/항체 희석액으로 교체하였다.
37℃/5% CO2에서 16 내지 24 h 동안 인큐베이션 후에, 100 ㎕ 브라이트글로(BrighiGlo) 루시퍼라제 시약 (프로메가 (Promega))을 첨가하고, 플레이트를 10 min 동안 인큐베이션하였다. 발광 판독 (테칸 플레이트 판독기)을 위해, 세포 용해물을 96웰 마이크로타이터 플레이트 (코스타 (Costar), Cat # 3917)로 옮겼다.
유사한 검정을 또한 Wnt3 또는 Wnt3a 과다-발현 세포 (예를 들어, CHO-K1, TM3, L 또는 HEK293 세포)의 동시 배양을 이용하여 수행할 수 있다.
(
ii
) 일시적으로 형질감염된 세포를 사용한
Wnt1
/
Wnt3a
리포터 유전자 검정
3x104개 HEK293T/17 세포/웰을 96웰 조직 배양 플레이트 (코스타) 내에 접종하고, 표 3에 기재된 바와 같이 세포를 37℃/5% CO2에서 100 ㎕ 배지 내에서 인큐베이션하였다. 12 내지 16 h 후에, 세포를 빈 벡터 Wnt 발현 플라스미드 1 ng/웰; pTA-Luc-10xSTF (반딧불이 (Firefly) 루시퍼라제 구축물) 50 ng/웰; 또는 phRL-SV40 (레닐라 (Renilla) 루시퍼라제 구축물) 0.5 ng/웰로 형질감염시켰다.
상기 나열된 플라스미드 및 0.2 ㎕ 퓨젠(FuGene6)6/웰 (로슈 (Roche))을 함유하는 형질감염 프리믹스 (premix) (10 ㎕/웰)를 제조하였다. 형질감염 프리믹스를 RT에서 15 min 동안 인큐베이션한 다음 웰 내로 분포시켰다. 플레이트를 400 rpm에서 2 min 동안 RT에서 흔든 후, 4 h 동안 37℃/5% CO2에서 인큐베이션하였다. 한편, 항체를 배지 내에 희석하고, 형질감염된 세포 (75 ㎕/웰)에 첨가하였다.
18 내지 24 h 후에, 75 ㎕/웰 듀얼글로 (DualGlo) 루시퍼라제 시약 (프로메가)을 첨가하고, 플레이트를 세포 용해를 위해 10 min 동안 흔든 후, 반딧불이 루시퍼라제 활성을 판독하였다. 발광 판독 후에, 75 ㎕/웰 듀얼글로 스탑&글로우(Stop&Glow) 시약 (프로메가)을 첨가하고, 레닐라 루시퍼라제 활성을 결정하기 위해 발광을 다시 측정하였다.
분석을 위해, 반딧불이 루시퍼라제 활성 및 레닐라 루시퍼라제 활성 사이의 비를 계산하였다. 항-LRP6 항체의 IC50-결정을 위해, 그래프패드 프리즘을 이용하여 상대적인 루시퍼라제 값을 분석하였다.
Wnt1 또는 다른 Wnt1 클래스 리간드 과다-발현 세포 (예를 들어 CHO-K1, TM3, L 또는 HEK293 세포)의 동시 배양을 이용하여 유사한 검정을 또한 수행할 수 있다.
9:
FACS
교차-반응성 연구
뮤린 및 시노몰구스 LRP6에 대한 교차-종 반응성을 FACS 분석에 의해 세포 상에서 결정하였다. FACS 염색을 본질적으로 상기 설명된 바와 같이 수행하였다. 인간 U266 세포 (LRP6의 발현 없이)를 음성 대조군으로서 사용하였다.
뮤린 LRP6에 대한 교차-반응성을 뮤린 NIH-3T3 세포 상에서 시험하였다. 시노몰구스 LRP6에 대한 교차-반응성을 시노몰구스 세포주 Cynom-K1 및 일시적으로 형질감염된 HEK293T/17 세포 상에서 시험하였다:
인간 세포주 HEK293T/17 상에서 시노몰구스 교차-반응성을 시험하기 위해, 세포를 제조자의 지시에 따라 리포펙타민 (Lipofectamine; 인비트로젠))을 사용하여 일시적으로 형질감염시켰다. 세포를 인간 LRP6 발현 플라스미드 pCMV6_XL4_LRP6 및 샤페론 (chaperone)-코딩 플라스미드 pcDNA3.1-flag_MESD의 혼합물 또는 pcDNA3.1-nV5-DEST_cynoLRP6 및 pcDNA3.1-flag_MESD의 혼합물 (시노몰구스 LRP6의 과다발현)로 형질감염시켰다. T175 플라스크 당 50 ㎍의 LRP6 발현 플라스미드 및 20 ㎍의 MESD 발현 플라스미드를 사용하였다. 24 h 후에, 세포를 탈착시키고, 염소 항 인간 LRP6 대조군 항체 (알앤디 시스템즈 (R&D Systems)) 및 항-LRP6 HuCAL 항체로 염색하였다. 음성 대조군 염색을 위해 모의-형질감염된 HEK293T/17 세포를 사용하였다 (낮은 내인성 LRP6 발현).
10: 결합제 최적화
친화도 성숙 라이브러리의 생성
선택된 항체 단편의 친화도 및 생물학적 활성을 증가시키기 위해, L-CDR3 및 HCDR2 영역을 트리뉴클레오티드 지정 돌연변이 유발을 이용하는 카세트 돌연변이 유발에 의해 동시에 최적화시켰고, 한편 프레임워크 영역을 일정하게 유지시켰다.
표준 클로닝 절차 및 전기-적격 이. 콜라이 TOP10F 세포 (인비트로젠) 내로 다양화된 클론의 형질전환에 의해 상이한 친화도 성숙 라이브러리를 생성하였다. 무작위하게 선발된 클론의 서열 결정은 100%의 다양성을 보여주었다. 선발한 클론들 사이에서 모 결합제는 발견되지 않았다. 마지막으로, 모든 라이브러리의 파지를 따로 제조하였다.
11:
MMTV
-
Wnt1
이종이식편
MMTV-Wnt1 트랜스제닉 마우스로부터의 종양을 종양 조각으로서 FVB 마우스의 유방 지방체 (fat pad) 내에서 5 계대배양 (passage) 동안 계대배양한 후, 누드 마우스의 유방 지방체 내로 이식하였다. 이식의 11일 후에, 종양이 약 110 mm3의 평균 부피에 도달했을 때, 마우스를 8마리 마우스/군의 3개의 군으로 무작위로 배정하고, 3일 마다 투약하였다 (DeAlmeida et al. (2007); Cancer Res. 67:5371-9).
12: 생화학적 검정에서 억제
HEK293 세포를 10% 소 태아 혈청을 보충한 D-MEM 내에서 37℃에서 5% CO2 하에 성장시켰다. 세포를 96웰 조직 배양 플레이트 (코스타) 내에 3x104/웰로 접종하고, 0.1 ng/웰 Wnt 발현 플라스미드, 50 ng/웰 STF 리포터, 및 0.2 ㎕/웰 퓨젠6 (로슈)와 혼합한 0.5 ng/ml phRL-SV40 (프로메가)로 형질감염시켰다. 형질감염 4시간 후에, 항체를 PBS 내에 희석하고, 형질감염된 세포에 첨가하였다. 18 h 인큐베이션 후에, 듀얼글로 루시퍼라제 시약 (프로메가)을 사용하여 반딧불이 루시퍼라제 및 레닐라 루시퍼라제 활성을 측정하였다. 형질감염 효율을 정규화하기 위해 레닐라 루시퍼라제를 사용하였다.
시험한 모든 IgG 포맷은 Wnt1 (2, 6, 7A, 7B, 9, 10A, 10B) 생성된 정형적 신호의 강력하고 완전한 억제 효과를 가진다. 이들은 모두 Wnt3/3A 생성된 신호의 존재 하에 종 (bell) 형상의 강화 곡선을 생성시킨다.
13:
FACS
-기반 경쟁 검정
FACS-기반 경쟁 검정을 위해, 제조자의 지시에 따라 ECL 단백질 비오티닐화 모듈 (지이 헬쓰케어)을 사용하여 항-LRP6 Fab 및 음성 대조군 Fab MOR03207를 비오티닐화하였다. 비오티닐화된 Fab를 일정 Fab 농도 (20 nM 최종 농도)에서 HEK293-hLRP6ΔC-eGFP 세포에 대한 FACS 염색을 위해 사용하고, 100배 몰 과량의 표지되지 않은 Fab와 경쟁시켰다. 세포를 Fab 희석액과 함께 1시간 동안 4℃에서 플레이트 진탕기 상에서 인큐베이션하였다. 세포를 FACS 완충제로 1x 세척한 후에, PE-접합된 스트렙타비딘 (디아노바 (Dianova))과 함께 1시간 동안 4℃에서 플레이트 진탕기 상에서 암소에서 인큐베이션하였다. 세포를 FACS 완충제로 2회 세척하고, FACS 어레이 (BD)를 사용하여 형광을 측정하였다.
유사하게, 비오티닐화되지 않은 항-LRP6 Fab를 100배 몰 과량의 LRP6 단백질 SOST와 경쟁시키고, 세포에 대한 Fab의 결합을 PE-접합된 항 인간 IgG 항체 (디아노바)에 의해 모니터링하였다.
14:
면역블로팅
검정
총 세포 용해물을 RIPA 완충제 (50 mM 트리스(Tris)-HCl, pH 7.4, 150 mM NaCl, 1% NP-40, 0.5% 나트륨 데옥시콜레이트, 0.1% SDS, 1 mM EDTA) 내에 제조하였다. 용해물을 단백질 농도에 대해 정규화하고, SDS-PAGE에 의해 분해하고, 니트로셀룰로스 막으로 옮기고, 지시된 항체로 프로빙하였다. pT1479 LRP6 항체는 만족스러운 결과를 위해 막 추출물의 생성을 요구한다. 막 추출물을 생성하기 위해, 4회 동결-해동 사이클을 수행함으로써 세포를 저장성 완충제 (10 mM 트리스-HCl pH 7.5, 10 mM KCl) 내에 용해시키고, RIPA 완충제를 사용하여 불용성 막 분획을 가용화시켰다. 프로테아제 억제제 칵테일 (시그마) 및 1x 포스파타제 억제제 칵테일 (업스테이트 (Upstate))을 용해 완충제에 첨가하였다. 웨스턴 블롯 검정에 사용된 시판 항체는 토끼 항-LRP6, 토끼 항-pT1479 LRP6, 및 토끼 항-pS1490 LRP6 항체 (셀 시그날링 테크놀로지 (Cell Signaling Technology))를 포함하였다.
실시예
1:
FACS
에 의한 내인성
LRP6
에 대한 항-
LRP6
항체의 특이적 결합
항-LRP6 항체 및 FACS 분석을 이용하여 많은 종양 세포 상에서 LRP6의 내인성 세포 표면 발현의 검출을 검사하였다. 도 1aa 및 1ab에 제시된 바와 같이, PA1 세포는 LRP5 및 LRP6 mRNA을 모두 발현하지만, U266 및 다우디 세포는 LRP6 mRNA를 발현하지 않는다. PA1 세포는 프로펠러 1 항-LRP6 IgG를 사용한 유의한 염색을 보이지만, U266 및 다우디 세포는 그렇지 않다. 보다 중요하게는, U266 세포는 항-LRP6 항체에 의해 염색되지 않지만, 이들은 LRP6을 발현하고, 이것은 항-LRP6 항체의 특이성을 입증한다. 또한, PA1 세포의 항-LRP6 항체 염색은 LRP6 shRNA를 사용한 내인성 LRP6의 결핍 시에 유의하게 감소하였고, 이것은 LRP6 항체의 특이성을 추가로 입증한다.
추가의 연구에서, PA1 세포에서 shRNA에 의한 LRP6의 녹다운은 LRP6에 대한 Prop1 및 Prop3 항체의 특이성을 추가로 확인한다 (도 1ba 및 1bb 참조). 녹다운은 세포를 LRP6에 대한 짧은 헤어핀 RNA를 코딩하는 렌티바이러스로 감염시키고 감염된 세포의 안정한 풀을 선택함으로써 달성하였다. 연구를 위해 사용된 shRNA 감염 방법은 문헌 [Wiederschain et al. in 2009 Cell Cycle 8: 498-504. Epub 2009 Feb 25]에 기재되어 있다.
실시예
2: 프로펠러 1 및 프로펠러 3 항-
LRP6
Fab
에 의한
Wnt1
및
Wnt3a
리포터 유전자 검정의 차별적 억제
상이한 항-LRP6 Fab를 시험관내 Wnt 리포터 검정으로 시험하였다. Wnt1 또는 Wnt3A 리간드를 HEK293T/17 STF 세포에서 일시적으로 발현시키고 (유전자 리포터 검정), 다양한 농도의 항-LRP6 Fab 단편으로 시험하였다. 문헌 [Huang et al. (2009), Nature; 461:614-20. Epub 2009 Sep 16]에 설명된 프로토콜을 이용하여 STF 검정을 수행하였다. 도 2a에 제시된 바와 같이, 프로펠러 1 항-LRP6 Fab (MOR08168, MOR08545, MOR06706)은 Wnt3A-의존성 신호 전달에 대한 많은 효과 없이 Wnt1-의존성 신호 전달을 특이적으로 감소시켰다. 이와 반대로, 도 2b에 제시된 바와 같이, 프로펠러 3 항-LRP6 Fab (MOR06475, MOR08193, MOR08473)은 Wnt1-의존성 Wnt1-의존성 신호 전달에 대한 유의한 효과 없이 Wnt3A-의존성 신호 전달을 특이적으로 감소시켰다. 결과는 Wnt1 및 Wnt3A 활성이 상이한 LRP6 Fab 단편 (에피토프)에 의해 따로 차단됨을 입증한다.
실시예
3: 상이한 종의
LRP6
에 대한 항-
LRP6
항체의 결합.
교차-반응성을 보여주기 위해, 인간 (HEK293T/17) 및 마우스 기원 (NIH 3T3)의 내인성 LRP6을 발현하는 세포, 또는 시노몰구스 LRP6을 발현하는 일시적으로 형질감염된 HEK293/T17 세포를 처리하고 상기 설명된 바와 같은 유동 세포측정법으로 처리하였다. 도 3은 발견의 결과를 요약하고, 모든 항-LRP6 항체가 인간, 마우스, 및 시노몰구스 LRP6에 결합함을 보여준다.
실시예
4: 프로펠러 1 및 프로펠러 3 항-
LRP6
항체에 대한 민감도에 기반한
Wnt
의 분류.
프로펠러 1 및 프로펠러 3 항-LRP6 항체에 대한 그들의 민감도에 기초하여 Wnt를 평가하기 위해, 다양한 Wnt 리간드를 HEK293T/17 STF 세포 내로 일시적으로 발현시키고 (유전자 리포터 검정), 프로펠러 1 또는 프로펠러 3 항-LRP6 항체로 처리하였다. 문헌 [Huang et al. (2009), Nature; 461:614-20. Epub 2009 Sep 16]에 설명된 프로토콜을 이용하여 STF 검정을 수행하였다. 결과를 LRP6의 특이적 프로펠러 영역에 대한 항체 결합/차단을 기초로 한 특정 Wnt의 활성 억제를 도시한 도 4에 제시한다. 도 4는 Wnt1, Wnt2, Wnt6, Wnt7A, Wnt7B, Wnt9, Wnt10A, Wnt10B에 의해 유도된 신호 전달은 프로펠러 1 항-LRP6 Fab에 의해 특이적으로 억제될 수 있는 한편, Wnt3 및 Wnt3A에 의해 유도된 신호 전달은 프로펠러 3 항-LRP6 Fab에 의해 특이적으로 억제될 수 있음을 보여준다.
실시예
5:
Fab
단편으로부터
IgG
로의
LRP6
항체의 전환은 다른 클래스의
Wnt
에 의해 유도된 Wnt 신호 전달을 강화한다.
다소 예기치 않은 관찰에서, Fab 단편으로부터 IgG로의 LRP6 항체의 전환은Wnt 신호 전달을 강화한다. 프로펠러 1 항-LRP6 IgG는 293T/17 STF 리포터 검정에서 Wnt1-의존성 신호 전달을 억제하고 Wnt3A-의존성 신호 전달을 강화한다. 유사하게, 프로펠러 3 항-LRP6 IgG는 도 5에 제시된 바와 같이 Wnt3A-의존성 신호 전달을 억제하고 Wnt1-의존성 신호 전달을 강화한다. 상기 발견은 Wnt 신호 전달 경로가 프로펠러 1 및 프로펠러 3 항체에 의해 변형되거나 또는 "미세 조정 (fine tuned)"될 수 있음을 제안한다. 다른 세포 배경 (예를 들어 MDA-MB231, MDA-MB435, PA-1, TM3 및 3t3 세포 - 데이터 미제시)에서 STF 리포터 검정에서 유사한 효과가 관찰되었다.
실시예
6: 프로펠러 1 또는 프로펠러 3 항-
LRP6
Fab
는
Wnt1
또는
Wnt3A
-유도된
LRP6
인산화를 특이적으로 억제한다.
HEK293T/17 세포를 Wnt1 또는 Wnt3A 발현 플라스미드로 일시적으로 형질감염시키고, 프로펠러 1 또는 프로펠러 3 항-LRP6 Fab로 처리하였다. 도 6에 제시된 바와 같이, 프로펠러 1 항-LRP6 Fab는 LRP6, 예를 들어,T1479 및 S1490 부위의 Wnt1-유도된 인산화를 특이적으로 억제하지만, 프로펠러 3 항체는 그렇지 않다. 이와 대조적으로, 프로펠러 3 항-LRP6은 LRP6의 Wnt3A-유도된 인산화를 특이적으로 억제하지만, 프로펠러 1 항체는 그렇지 않다. 상기 결과는 항체들이 LRP6의 구분되는 프로펠러 도메인에 결합하고 특이적 Wnt 리간드를 차단함을 지지한다.
실시예
7: 프로펠러 1 항-
LRP6
IgG
를 사용한
MMTV
-
Wnt1
종양 이종이식편 모델에서
Wnt
표적 유전자의 발현의 억제.
LRP6 항체를 MMTV-Wnt1로서 공지된 당업계에서 인정되는 유전공학 처리된 마우스 모델에서 생체 내에서 추가로 특성 결정하였다. IgG 포맷의 항-LRP6 항체가 생체 내에서 Wnt 신호 전달 및 종양 성장을 억제할 수 있는지 결정하기 위해 실험을 수행하였다. MMTV-Wnt1 트랜스제닉 마우스로부터 유래한 유방 종양은 Wnt1 의존성이고; 테트라사이클린-조절된 시스템을 사용하여 Wnt1 발현을 끄거나 (Gunther et al. (2003), 상기 문헌), 또는 Fz8CRDFc를 사용하여 Wnt 활성을 차단하면 (DeAlmeida et al. (2007) Cancer Research 67:5371-5379) 생체 내에서 종양 성장을 억제한다. MMTV-Wnt1 종양에서 Wnt 신호 전달에 대한 항-LRP6 항체의 효과를 측정하기 위해, MMTV-Wnt1 종양을 이식한 마우스에게 단일 용량의 5 mg/kg Wnt1 클래스-특이적 길항작용성 항-LRP6 항체를 i.v. 투여하였다. 항체의 혈청 농도 및 β-카테닌 표적 유전자 액신2의 mRNA 발현을 2주 기간에 걸쳐 분석하였다. LRP6 항체의 말기 β-기 반감기는 약 108시간이었다. 항체 주사에 상응하여, 종양에서 액신2 mRNA 발현의 유의한 감소가 관찰되었고, 액신2 발현은 항체 주사 후 1주에 점차 회복하였고, 이때 혈청 내 항체 수준은 감소하였다. 이들 결과는 Wnt1 클래스-특이적 항-LRP6 항체가 MMTV-Wnt1 이종이식편에서 Wnt 신호 전달을 억제함을 제안하고, 상기 제안은 혈청 내의 LRP6 항체의 농도와 상관성이 있다. 종양 성장에 대한 항-LRP6 항체의 효과를 시험하기 위해, 마우스에게 프로펠러 1-, 프로펠러 3-특이적 항-LRP6 항체, 또는 이소형 매칭된 대조군 항체를 투여하였다. 마우스에게 20 mg/kg의 초기 용량을 i.v. 투여한 후, 10 mg/kg를 3일마다 투여하였다. 상기 실험에서, 프로펠러 1-특이적 항-LRP6 항체는 종양 퇴행을 유발하지만 프로펠러 3-특이적 항-LRP6 항체는 그렇지 않았다. 함께 살펴보면, 이들 결과는 프로펠러 1-특이적 항-LRP6 항체가 MMTV-Wnt1 이종이식편의 퇴행을 유도함을 입증한다.
누드 마우스에서 MMTV-Wnt1 종양 이종이식편을 1시간 내지 14일 범위의 상이한 시점에 MOR08168 (프로펠러 1 항-LRP6 IgG)로 처리하였다. 도 7은 항체의 PK 혈청 농도 및 Wnt 표적 유전자 액신2의 mRNA 발현과의 역상관관계를 보여준다. 종양에서 액신2 유전자 발현 수준은 MOR08168 처리로 억제되고, 항체가 혈청으로부터 청소될 때 회복한다.
액신2에 추가로, 추가의 유전자의 발현에 대한 MOR08168의 효과를 평가하였다. 아피메트릭스 (Affymetrix) 마우스(Mouse)430 2.0 어레이를 사용하여, MMTV-Wnt1 동종이식편 종양 + 또는 - 단일 용량의 MOR08168 (5 mg/kg)의 실험의 시간 경과를 프로파일링하였다. 모두 6개의 시점 (0, 1, 3, 8, 24, 336시간)이 있고, 시점당 3개의 반복 실험을 하였다. 항체를 사용한 처리 시에 액신의 최대 억제를 입증하는 데이터에 기반하여, Wnt 경로 억제에 추정적으로 반응하는 차별적 발현된 유전자를 결정하기 위해 최상의 대표적인 시점으로서 8 h을 선택하였다. R/바이오컨덕터 (Bioconductor) 프레임워크를 이용하고, Limma 패키지를 사용하여 0시간 시점 내지 8시간 시점 사이에서 차별적 발현된 유전자를 결정하였다. 차별적 발현된 유전자의 세트를 결정하기 위해 .05의 조정된 P-값을 역치로서 사용하였다. 상기 컷오프 (cutoff)에 기초하여, 972 유전자(들)에 차별적 발현된 맵핑된 것으로 불리는 1270개의 프로브 세트가 존재한다. 도 8aa-8ac는 <0.01의 조정된 P-값에서 >2배 상향조절된 유전자를 보여주는 표이고, 도 8ba-8be는 <0.01의 조정된 P-값에서 >2배 하향조절된 유전자를 보여주는 표이다.
실시예
8:
MMTV
-
Wnt1
동종이식편
모델에서 프로펠러 1 및 프로펠러 3 항-
LRP6
항체의 항-종양 활성.
LRP6 프로펠러 1 및 3 항체의 항-종양 활성을 MMTV-Wnt1 동종이식편 모델에서 평가하였다. MMTV-Wnt1 종양 단편을 암컷 누드 마우스에 피하 (s.c.) 이식하였다. 이식 11일 후에, MMTV-Wnt1 종양을 보유한 마우스 (n=8, 평균 121 mm3; 범위: 100-147 mm3)를 비히클 IgG (10 mg/kg, 정맥내 (i.v.), 3일마다 (q3d)), LRP6-프로펠러 1 Ab MOR08168 (10 mg/kg, i.v., q3d), 또는 LRP6-프로펠러 3 Ab MOR06475 (10 mg/kg, i.v., q3d)으로 처리하고, 종양을 3일마다 캘리퍼스로 측정하였다. LRP6-프로펠러 1 MOR08168 Ab는 종양 퇴행을 용량-의존적으로 유도하였다 (-55%, p<0.05) (도 9 참조).
실시예
9:
MMTV
-
Wnt3
동종이식편
모델에서 프로펠러 1 및 프로펠러 3 항-
LRP6
항체의 항-종양 활성.
LRP6 프로펠러 1 및 3 항체의 항-종양 활성을 MMTV-Wnt3 동종이식편 모델에서 평가하였다. MMTV-Wnt3 종양 단편을 암컷 누드 마우스에 피하 (s.c.) 이식하였다. 이식 15일 후에, MMTV-Wnt3 종양을 보유한 마우스 (n=6, 평균 209 mm3; 범위: 113-337 mm3)를 비히클 IgG (10 mg/kg, 정맥내 (i.v.), 매주 2회 (2qw)), MOR08168 LRP6-프로펠러 1 Ab (3 mg/kg, i.v., qw), 또는 MOR06475 LRP6-프로펠러 3 Ab (10 mg/kg, i.v., 2qw)로 처리하고, 종양을 매주 2회 캘리퍼스로 측정하였다. MOR06475 LRP6-프로펠러 3 Ab는 항종양 활성을 나타냈다 (T/C=34%, p<0.05) (도 10 참조).
실시예
10: 생체 내에서
Wnt3A
를 억제하는 프로펠러 1 및 3 항-
LRP6
항체의 능력의 평가
생체 내에서 Wnt3 클래스 Wnt 신호 전달을 억제하는 LRP6-프로펠러 1 및 3 항체의 능력을 Wnt3A 분비 L 세포를 동시-이식한 PA1-STF 리포터 세포로 이루어지는 동시-이식편 시스템에서 시험하였다. 암컷 누드 마우스에게 10x10e6 PA1-STF 세포 및 0.5x10e6 L-Wnt3A 세포를 피하 이식하고, 5개의 군에 무작위로 배정하였다. 24시간 후에, 마우스에게 비히클, MOR08168 LRP6-프로펠러 1 Ab (10 mg/kg), 또는 MOR06475 LRP6-프로펠러 3 Ab (10 mg/kg)의 단일 정맥내 용량을 투여하고, 6시간, 24시간, 48시간, 72시간 및 168시간 후에 제노겐 (Xenogen)에 의해 영상화하였다.
MOR06475는 적어도 72시간 동안 Wnt3a 유도된 신호 전달을 억제할 수 있는 반면, Mor08168은 Wnt3A 유도된 신호 전달을 증가시켰다 (도 11).
실시예
11:
HDx
MS
에 의한
LRP6
PD3
/4 및 그의 항체 복합체의
에피토프
맵핑
프로펠러 3의 YWTD-EGF 영역 내에서 항체 결합 부위를 확인하기 위해, 수소-중수소 교환 (HDx) 질량 분광법 (MS)을 사용하였다. LRP6 프로펠러 도메인 3-4 (PD3/4)는 12개의 시스테인 및 4개의 N-연결된 글리코실화 부위를 가졌다. 4개의 모든 N-연결된 글리코실화 부위는 프로펠러 3 도메인 (631-932) 내에 위치한다. HDx MS에 의해, 100%에 가까운 적용범위 (그에 대한 구조적 정보를 얻기 위해 가능한 단백질의 양)는 프로펠러 4 도메인 상에 맵핑되고, 약 70% 적용범위는 프로펠러 3 도메인 상에 맵핑되었다. 4개의 글리코실화된 부위를 바로 둘러싸는 영역은 검출되지 않고 남는다 (도 12a). Fab 06745의 존재 하에, 2개의 약하게 용매 보호된 펩티드가 프로펠러 3 (Phe636-Leu647, Tyr844-Glu856; 도 11A)에서 발견되었다. 이것은 에피토프를 담당하는 잔기 또는 잔기의 분획이 보호된 펩티드 상에 또는 공간적으로 인근에 존재함을 제안한다. LRP6 PD34의 결정 구조에 기초하여, 용매 보호된 영역은 Prop 3의 날개깃 1과 6 사이의 오목한 표면에 대응한다.
ScFv06475
와
LRP6
PD34
와의 상호작용을 파괴하는 돌연변이
날개깃 1 및 6의 테두리가 Wnt3a 신호 전달의 항체 매개된 억제를 담당하는 것을 확인하기 위해, HDx에 의해 결합에 연루된 영역을 또한 대략 덮는 일련의 LRP6 표면 돌연변이를 제작하였다 (R638A, W767A, Y706A/E708A, W850A/S851A, R852/R853A, 및 D874A/Y875A). 돌연변이체 단백질이 적합하게 폴딩되는 것을 보장하기 위해, 차별적 정적 광 산란 (DSLS) 열-용융 검정을 수행하였다. 온도 변성 실험은 야생형 및 돌연변이체 단백질의 응집 온도 Tagg (50%의 단백질이 변성되는 온도)가 유사함을 보여주었다. 따라서, 돌연변이는 단백질의 폴딩 또는 안정성에 대해 효과가 없었다.
scFv MOR06475에 대한 돌연변이체 LRP6의 결합 능력을 ELISA에 결정하였다. HDx MS 실험에서 용매 보호를 보여준 펩티드 상에 위치하는 잔기 (R638, W850/S851, 및 R852/R853)의 돌연변이는 또한 항체 결합의 극적인 감소를 보여주었다 (도 12b 참조). HDx에서 용매 보호를 보이지 않는 펩티드 상에 위치하는 잔기 (Y706/E708)의 돌연변이는 또한 항체 결합 능력에서 변화를 보이지 않았다 (도 11B). 따라서, 결합 검정 데이터는 잔기 R638, W850, S851, R852, 및 R853이 에피토프에 직접 참여하는 것을 제안하는 HDx MS에 의해 맵핑될 때 결합 계면과 우수하게 일치한다.
종합하면, 이들 결과는 상이한 Wnt 단백질이 신호 전달을 위해 LRP6의 상이한 프로펠러 영역을 요구함을 보여준다. Wnt1 클래스의 Wnt 단백질 (Wnt1, 1, 2, 6, 7A, 7B, 9, 10A, 10B)은 Wnt1 신호 전달을 위해 LRP6의 프로펠러 1을 요구하고, 이들은 프로펠러 1 특이적 항-LRP6 항체에 의해 억제될 수 있다. Wnt3A 클래스의 Wnt 단백질 (Wnt3 및 Wnt3a)은 Wnt3 신호 전달을 위해 LRP6의 프로펠러 3을 요구하고, 이들은 프로펠러 3 특이적 항-LRP6 항체에 의해 억제될 수 있다 (도 13). 또 다른 예기치 않은 결과는 Wnt 리간드의 존재 하에 이가 IgG 포맷의 항체의 Wnt-강화 활성이었다. IgG 포맷의 시험한 모든 항체는 STF Luc 리포터 유전자 검정에서 Wnt1 또는 Wnt3A 신호 전달을 향상시켰다. 흥미롭게도, Wnt1을 억제하고 Wnt3A 검정에서 불활성인 대부분의 Fab는 IgG로서 여전히 Wnt1을 억제하였지만, Wnt3A 신호 전달을 강화하고 그 반대도 마찬가지이다. Wnt3A를 억제한 대부분의 Fab는 IgG로서 Wnt1 활성을 강화하였다. 몇몇 포맷을 시험할 때 (IgG1LALA, IgG2, IgG4, IgG4_Pro), 효과는 IgG 포맷에 독립적이었다. 이들 데이터는 상이한 Wnt 단백질이 신호 전달을 위해 LRP6의 상이한 프로펠러에 결합하는 것을 보여준다. 이가 LRP6 항체를 사용한 LRP6의 이량체화는 홀로 Wnt 신호 전달을 자극하기에 충분하지 않고, 대신에 다른 클래스의 Wnt 단백질에 의해 개시된 Wnt 신호 전달을 강화할 수 있다. 이들 발견은 상이한 정형적 Wnt 리간드는 LRP6 상의 구분되는 결합 부위를 이용하고, 모든 이가 항체는 비-차단된 Wnt Fab (단일 쇄 항체, 유니바디)의 존재 하에 IgG 포맷에서 Wnt 활성을 향상시킴을 입증한다. 최종 포맷으로서 임의의 종류의 일가 구조는 Wnt 강화를 방지한다. 별법으로, Wnt1- 및 Wnt3A 억제성 활성을 모두 보유하는 이중특이적 IgG 또는 IgG-유사 분자는 강화를 방지한다. 따라서, LRP6 항체 구축물은 Wnt 경로를 제어하고 "미세-조정"하도록 설계될 수 있다.
실시예
12:
이중파라토프
LRP6
항체의 생성
본 실시예는 이중파라토프 항-LRP6 IgG-scFv 항체의 생산 및 특성 결정을 설명한다. 상이한 도메인 배향 (예를 들어 VH-VL 또는 VL-VH) 및 상이한 링커 길이 (예를 들어, (Gly4Ser)3 또는 (Gly4Ser)4)를 함유하는 다양한 항-LRP6 scFv을 초기에 발현시키고, 정제하고, 특성 결정하였다. scFv 연구의 결과에 기초하여, 상이한 이중파라토프 항-LRP6 IgG-scFv를 제조하고, 추가로 평가하였다. scFv는 CH3 또는 CL의 C-말단 및 VH 또는 VL의 N-말단을 비롯한 IgG 내의 다양한 위치에 위치할 수 있다. 또한, Gly4Ser 및 (GlyGlySer)2를 비롯한, scFv를 IgG에 연결하기 위해 다양한 링커를 사용할 수 있다.
(a) 물질 및 방법
(i) 항-
LRP6
scFv
의 생성
모든 scFv-변이체를 코딩하는 유전자를 진아트 (Geneart)에 의해 합성하였다. 두 배향으로 scFv를 코딩하는 DNA-단편 (VH-VL 및 VL-VH, 2개의 상이한 링커: (Gly4Ser)3 및 (Gly4Ser)4에 의해 분리됨, N-말단 신호 서열 및 C-말단 6xHis-태그를 가짐)을 진아트 벡터로부터 NdeI/XbaI를 통해 벡터 pFAB15-FkpA 내로 직접 클로닝하였으며, 생성되는 구축물은 pFab15-MOR06475-VH-(Gly4Ser)3-VL, pFab15-MOR06475-VH-(Gly4Ser)4-VL, pFab15-MOR06475-VL-(Gly4Ser)3-VH, pFab15-MOR06475-VL-(Gly4Ser)4-VH, pFab15-MOR08168-VH-(Gly4Ser)3-VL, pFab15-MOR08168-VH-(Gly4Ser)4-VL, pFab15-MOR08168-VL-(Gly4Ser)3-VH, pFab15-MOR08168-VL-(Gly4Ser)4-VH, pFab15-MOR08545-VH-(Gly4Ser)3-VL, pFab15-MOR08545-VH-(Gly4Ser)4-VL, pFab15-MOR08545-VL-(Gly4Ser)3-VH 및 pFab15-MOR08545-VL-(Gly4Ser)4-VH로 불렸다.
(ii) 이중파라토프 항-LRP6 IgG-scFv의 생성
항-
LRP6
_
MOR08168
hIgG1LALA
_6475
scFv
코돈-최적화된 VH-서열 (진아트에 의해 합성된)을 함유하는 벡터 pRS5a MOR08168 hIgG1LALA를 이중파라토프 구축물의 생성을 위한 공급원으로서 사용하였다. 초기에, AfeI-부위를 hIgG1LALA를 코딩하는 서열의 3'-말단에서 퀵체인지 (QuickChange) 부위-지정 돌연변이 유발 (스트라타젠 (Stratagene))에 의해 도입하였다 (프라이머 no. 1: (서열 213) 및 프라이머 no. 2: (서열 214)). MOR06475scFv를 코딩하는 유전자 (VL-VH-배향, (Gly4Ser)4-링커에 의해 분리됨)를 진아트에 의해 합성하였다. AfeI-부위를 함유하는 5'-프라이머 및 (GlyGlySer)2-링커를 코딩하는 서열뿐만 아니라 AscI-부위를 함유하는 3'-프라이머를 전체 MOR06475-scFv-유전자의 증폭을 위해 사용하였다 (프라이머 no. 3:
(서열 215) 및 프라이머 no. 4: (서열 216)). 이어서, PCR-생성물을 AfeI/AscI를 통해 벡터 pRS5a MOR08168 hIgG1LALA 내로 클로닝하여, 최종 구축물 pRS5a MOR08168 hIgG1LALA-6475sc-fv를 생성하였다. MOR08168의 코돈-최적화된 VL을 코딩하는 유전자를 진아트에 의해 합성하였다. AgeI-부위를 함유하는 5'-프라이머 및 HindIII-부위를 함유하는 3'-프라이머를 전체 MOR08168-VL-유전자의 증폭을 위해 사용하였다 (프라이머 no. 5: (서열 217) 및 프라이머 no. 6: (서열 218)). 이어서, PCR-생성물을 AgeI/HindIII을 통해 벡터 pRS5a-h람다 내로 클로닝하여, 최종 구축물 pRS5a MOR08168 h람다를 생성하였다.
Lys
(K)가 없는 항-
LRP6
_
MOR08168
hIgG1LALA
_6475
scFv
벡터 pRS5a MOR08168 hIgG1LALA-6475scFv (서열 165)를 CH3에서 C-말단 리신의 제거를 위해 주형 DNA로서 사용하였다. 퀵 체인지 XL 부위-지정 돌연변이 유발 키트 (스트라타젠)를 다음 프라이머와 조합으로 사용하였다:
부위-지정 돌연변이 유발은 스트라타젠 프로토콜에 따라 이루어졌고; 생성되는 새로운 구축물은 pRS5a hIgG1LALA MOR08168opt 6475 scFv K로 불린다.
코돈-최적화된 VL-서열 (진아트에 의해 합성된)을 함유하는 벡터 pRS5a MOR08168 h람다 (항-LRP6_MOR08168 hIgG1LALA_6475scFv에 대해 설명된 생성)를 LC의 발현을 위한 임의의 변형과 함께 또는 그 없이 사용하였다.
항-
LRP6
_
MOR08168
hIgG1LALA
_6475
scFv
_
AspPro
에서
AspAla
로 (
DP
에서
DA
로)
벡터 pRS5a MOR08168 hIgG1LALA-6475scFv (서열 165)를 scFv의 VH에서 DP의 DA로의 치환을 위한 주형 DNA로서 사용하였다. 퀵 체인지 XL 부위-지정 돌연변이 유발 키트 (스트라타젠)를 다음 프라이머와 조합으로 사용하였다:
부위-지정 돌연변이 유발은 스트라타젠 프로토콜에 따라 이루어졌고; 생성되는 새로운 구축물은 pRS5a hIgG1LALA MOR08168opt 6475 scFv DP에서 DA로 불렸다.
코돈-최적화된 VL-서열 (진아트에 의해 합성된)을 함유하는 벡터 pRS5a MOR08168 h람다 (항-LRP6_MOR08168 hIgG1LALA_6475scFv에 대해 설명된 생성)를 LC의 발현을 위한 임의의 변형과 함께 또는 그 없이 사용하였다.
항-
LRP6
_
MOR08168
hIgG1LALA
_6475
scFv
_
AspPro
에서
ThrAla
로 (
DP
에서
TA
로)
벡터 pRS5a MOR08168 hIgG1LALA-6475scFv (서열 165)를 scFv의 VH에서 DP의 TA로의 치환을 위한 주형 DNA로서 사용하였다. 퀵 체인지 XL 부위-지정 돌연변이 유발 키트 (스트라타젠)를 다음 프라이머와 조합으로 사용하였다:
부위-지정 돌연변이 유발은 스트라타젠 프로토콜에 따라 이루어졌고; 생성되는 새로운 구축물은 pRS5a hIgG1LALA MOR08168opt 6475 scFv DP에서 TA로 불렸다.
코돈-최적화된 VL-서열 (진아트에 의해 합성된)을 함유하는 벡터 pRS5a MOR08168 h람다 (항-LRP6_MOR08168 hIgG1LALA_6475scFv에 대해 설명된 생성)를 LC의 발현을 위한 임의의 변형과 함께 또는 그 없이 사용하였다.
ValLeu
(
VL
)에서 항-
LRP6
_
MOR08168hIgG1LALA
_6475
scFv
코돈-최적화된 VH-서열 (진아트에 의해 합성된)을 함유하는 벡터 pRS5a MOR08168 hIgG1LALA를 HC의 발현을 위한 임의의 변형과 함께 또는 그 없이 사용하였다.
코돈-최적화된 MOR06475scFv 및 MOR08168-VL을 코딩하는 유전자를 DNA2.0에 의해 합성하고, AgeI/HindIII을 통해 벡터 pRS5a h람다 MOR08168 내로 클로닝하였다. 생성되는 벡터는 VL에서 pRS5a h람다 MOR08168 6475scFv로 불렸다.
항- LRP6 _ MOR06475hIgG1 LALA _8168 scfv ( VH -3- VL ) (여기서, 3은 VH 및 VL 사슬 사이의 ( Gly 4 Ser ) 3 아미노산 링커를 나타낸다).
MOR06475-VH를 다음 프라이머를 사용하여 벡터 pM2 hIgG1LALA MOR06475로부터 증폭하였다:
PCR-생성물을 NruI/BlpI를 통해 벡터 pRS5a hIgG1LALA MOR08168 6475scFv 내로 클로닝하였고, 생성되는 벡터는 pRS5a hIgG1LALA MOR06475 6475scFv로 불렸다.
MOR08168scFv를 다음 프라이머를 사용하여 벡터 pRS5a MOR08168 scFv (VH-3-VL)로부터 증폭하였다:
이어서, PCR-생성물을 AfeI/XbaI를 통해 벡터 pRS5a hIgG1LALA MOR06475 6475scFv 내로 클로닝하였고, 생성되는 최종 벡터는 pRS5a hIgG1LALA MOR06475 8168scFv (VH-3-VL)로 불렸다.
MOR06475-VL을 다음 프라이머를 사용하여 벡터 pM2 h카파 MOR06475로부터 증폭하였다:
PCR-생성물을 AgeI/BsiWI를 통해 벡터 pRS5a h카파 MOR06654 내로 클로닝하였고, 생성되는 벡터는 pRS5a h카파 MOR06475로 불렸다.
항- LRP6 _ MOR06475hIgG1 LALA _8168 scfv ( VH -4- VL ) (여기서, 4는 VH 및 VL 사슬 사이의 ( Gly 4 Ser ) 4 아미노산 링커를 나타낸다).
MOR06475-VH를 다음 프라이머를 사용하여 벡터 pM2 hIgG1LALA MOR06475로부터 증폭하였다:
PCR-생성물을 NruI/BlpI을 통해 벡터 pRS5a hIgG1LALA MOR08168 6475scFv 내로 클로닝하였고, 생성되는 벡터는 pRS5a hIgG1LALA MOR06475 6475scFv로 불렸다.
MOR08168scFv를 다음 프라이머를 사용하여 벡터 pRS5a MOR08168 scFv (VH-4-VL)으로부터 증폭하였다:
MOR06475-VL을 다음 프라이머를 사용하여 벡터 pM2 h카파 MOR06475로부터 증폭하였다:
PCR-생성물을 AgeI/BsiWI를 통해 벡터 pRS5a h카파 MOR06654 내로 클로닝하였고, 생성되는 벡터는 pRS5a h카파 MOR06475로 불렸다.
(
iii
) 항-
LRP6
-
scFv
의 발현
전기-전능성 이. 콜라이 균주 W3110을 플라스미드-DNA로 형질전환시켰다. 예비-배양액 (500 ml 플라스크 내의 12.5 ㎍/ml 테트라사이클린 및 0.4% 글루코스를 함유하는 150 ml LB-배지)을 단일 콜로니로 접종하고, 37℃/230 rpm에서 밤새 인큐베이션하였다. 발현 배양액 (2 리터 플라스크 내의 12.5 ㎍/ml 테트라사이클린을 함유하는 6x500 ml SB-배지)을 0.1의 O.D600으로 예비-배양액으로 접종하고, 25℃/230 rpm에서 약 0.6의 O.D600로 인큐베이션하였다. 이어서, IPTG (로슈)를 0.4 mM의 최종 농도로 첨가하고, 배양액을 25℃/230 rpm에서 밤새 인큐베이션하였다. 세포를 원심분리 (4600 rpm, 4℃에서 20 min)에 의해 수거하고, 세포 펠릿을 -20℃에서 동결시켰다.
(
iv
) 항-
LRP6
-
scFv
의 정제
3 리터 발현액으로부터의 펠릿을 50 ml 용해-완충제 (20 mM NaH2PO4, 20 mM 이미다졸, 500 mM NaCl, pH 7.4; 50 ml 완충제당 EDTA가 없는 1 정제 컴플리트 (Complete), 로슈 # 11836170001, 10 mM MgSO4 및 벤조나제) 내에 현탁시켰다. 세포 현탁액을 프렌치 프레스 (French Press) (1000 bar에서 2x)로 처리하고, 30 min 동안 16000xg, 4℃에서 원심분리하였다. 1 ml HisTrap HP 칼럼 (지이 헬쓰케어 (GE Helthcare))을 10 ml 용해-완충제로 평형화시켰다. 상청액을 스테리컵 (Stericup) 필터 (밀리포어 (Millipore))를 통해 여과하고, 평형화된 칼럼 (1 ml/min) 내로 로딩하였다. 칼럼을 용해-완충제 (20 ml)로 세척하고, 결합된 단백질을 3 ml 용리-완충제 (용해-완충제와 같지만 250 mM 이미다졸 함유)로 용리하였다. 용리액을 수퍼덱스75-칼럼 (하이로드(HiLoad) 16/60, 지이 헬쓰케어) 상에 바로 로딩하고, 130 ml PBS (1 ml/min)로 평형화화였다. 1 ml/min에서 PBS를 사용하여 실행을 실시하고, 용리액을 1.5 ml 분획으로 수집하고, 10% 비스-트리스-겔(Bis-Tris-Gel) (누페이지(NuPage), 인비트로젠) 상에서 분석하였다. 적당한 분획을 모으고, 0.2 ㎛ 필터를 통해 여과하고, 4℃에서 저장하였다. 모든 정제된 단백질을 LC-MS (산화 및 환원된 샘플 사용) 및 SEC-MALS (응집 분석)에 의해 분석하였다.
(v)
이중파라토프
항-
LRP6
IgG
-
scFv
의 일시적 발현
3.2 리터의 HEK293-6E 세포를 Rocks 10 rpm, 각도 7도, 폭기 25 L/h, O2 25%, CO2 6%의 바이오웨이브(BioWave)20에서 2E6 생존가능 세포/ml의 밀도로 M11V3 배지: Lot# D07668B에서 배양하였다. 세포를 일시적으로 1.8 리터의 DNA:PEI-MIX (플라스미드: pRS5a MOR08168 hIgG1 LALA-6475sc-Fv 5 mg + pRS5a MOR08168 h람다 5 mg + 20 mg PEI)로 형질감염시켰다. 형질감염 6시간 후에, 이스톨레이트: Lot# 09-021가 있는 5 리터의 공급 배지 (노파르티스)를 배양액에 첨가하였다. 이어서, 세포를 록스(Rocks) 24 rpm, 각도: 7도, 폭기 25 L/h, O2 25%, CO2 0-6%에서 추가로 배양하였다. 형질감염 7일 후에, 프레세니우스 (Fresenius) 필터 (0.2 ㎛)를 사용하여 십자류 여과에 의해 세포를 제거하였다. 그 후, 세포 미함유 물질을 프레세니우스의 10 kDa 컷오프 필터를 사용하여 십자류 여과로 1.75 L로 농축하였다. 농축 후에, 농축액을 스테리컵 필터 (0.22 ㎛)를 통해 멸균 여과하였다. 멸균 상청액을 4℃에서 저장하였다. 모든 설명된 이중파라토프 항-LRP6-scFv 변이체를 유사한 방식으로 발현시켰다.
(
vi
)
이중파라토프
항-
LRP6
IgG
-
scFv
의 정제
이중파라토프 IgG의 정제는 25 ml의 자가충전된 맵셀렉트 슈어 수지 (모두 지이 헬쓰케어)가 있는 새로 위생처리된 (0.2 M NaOH/30% 이소프로판올) XK16/20 칼럼을 사용하여 냉각 캐비넷에서 6℃에서 AKTA 100 익스플로러 (explorer) 공기 크로마토그래피 시스템에서 수행하였다. 모든 유량은 5 bar의 압력 한계에서, 로딩을 제외하고 3.5 ml/min이었다. 칼럼을 3 CV의 PBS (10x로부터 제조, 깁코 (Gibco))로 평형화한 후, 농축시키고, 멸균 여과된 발효 상청액 (1.35 L)을 2.0 ml/min o/n으로 로딩하였다. 칼럼을 8 CV의 PBS로 세척하였다. 이어서, IgG를 50 mM 시트레이트, 70 mM NaCl, pH 4.5에서 출발하여 12 CV에서 50 mM 시트레이트, 70 mM NaCl, pH 2.5로 선형으로 감소시킨 후, 동일한 pH 2.5 완충제의 2 CV 일정 단계가 이어지는 pH 구배를 사용하여 용리하였다. 이중파라토프 IgG는 pH 3.8 근처에서 단일 대칭 피크에서 구배 동안 용리되었고, 4 ml 분획으로 수거하였다. 분획을 3개의 풀 (pool), 즉 좌측 경사 (slope), 주 피크 및 우측 경사로 모았다. 풀을 2 M 트리스, pH 9.0를 사용하여 서서히 교반하면서 pH 7.0로 즉시 적정하였다. 풀을 멸균 여과하고 (밀리포어 스테리플립 (Steriflip), 0.22 ㎛), OD 280 nm를 람다 35 분광기 (퍼킨 엘머 (Perkin Elmer))로 측정하고, 단백질 농도를 서열 데이터를 기초로 하여 계산하였다. 풀을 응집 (SEC-MALS) 및 순도 (SDS-PAGE 및 MS)에 대해 별개로 시험하고, 그 결과를 기초로 하여, 중앙의 풀 (2.55 mg/ml 단백질의 35 ml)만을 추가로 사용하였다. 모든 설명된 이중파라토프 항-LRP6-scFv 변이체를 유사한 방식으로 정제하였다.
(
vii
) 단백질
마이크로어레이에
의한 교차-반응성 분석
마이크로어레이는 프로타겐 아게 (Protagen AG) (유니칩(UNIchip)® AV-VAR-EP)에 의해 제조된 주문 제작한 고-밀도 단백질 칩이고, 니트로셀룰로스 코팅된 유리 슬라이드 상에 4중으로 인쇄된 384개의 예비 규정되고 정제된 인간 단백질을 함유하였다. 단백질은 유전자 실체를 기초로 하여 세포외 또는 분비 단백질로 분류되고, 에스케리키아 콜라이 (Escherichia coli)를 사용하여 N-말단 His-태그 융합 단백질로서 발현되고, 고정된 금속 이온 친화도 크로마토그래피 (IMAC)를 이용하여 정제하였다. 항체의 혼성화는 프로타겐 아게에서 개발된 프로토콜로 프로그래밍된 테칸 혼성화 스테이션 HS400Pro을 사용하여 이루어졌다. 1차 항체는 5 ㎍/ml의 최종 농도에서 시험하였다. 2차 표지 항체인 Cy5-접합된 AffiniPure 염소 항-hsIgG F(ab')2, 단편 특이적 (잭슨 이뮤노리써치 (Jackson Immunoresearch), 코드 번호. 109-175-097)을 7.5 ㎍/ml의 최종 농도로 사용하였다. 마이크로어레이 영상은 적색 레이저 (635 nm)가 장착된 진픽스 프로페셔널 (GenePix Professional) 4200A 형광 마이크로어레이 스캐너 (액손 인스트루먼츠 (Axon Instruments, 미국 캘리포니아주))를 사용하여 획득하였다. 진픽스 프로 (GenePix Pro) v6.0 소프트웨어를 사용하여 영상 분석을 수행하였다. 데이터 분석은 프로타겐 유니칩® 데이터 분석 툴 v1.8을 사용하여 수행하였다. 인쇄된 항원에 대한 2차 항체의 직접 결합으로부터 유래한 비특이적 교차-반응성을 결정하기 위해, 2차 항체를 또한 1차 항체를 사용하지 않으면서 유니칩® 상에서 인큐베이션하였다. 상응하는 항원에 대해 정규화된 교차반응성 수준의 결정을 위해, 포화 농도 (20 fmol/스팟 (spot))에서의 형광 신호 강도 값을 100%로 설정하였다. 항원 신호의 4%를 초과하는, 주어진 단백질에 대한 모든 신호 강도는, 그 강도가 2차 표지된 항체만을 사용하여 각각의 대조군에서 발견되지 않으면 양성 적중치 (hit)로 간주하였다.
(
viii
) 시차 주사 열량분석 (
DSC
)에 의해 측정된
입태형태적
안정성
DSC는 딥 (deep)-웰 플레이트 자동 샘플 채취기가 장착된, 마이크로칼 (Microcal)의 모세혈관 세포 마이크로열량계 VP-DSC를 사용하여 측정하였다. 데이터는 소프트웨어 오리진 (Origin) 7.5로 분석하였다. 샘플 (400 ㎕)을 2 ml 딥-웰 플레이트 (눈크 (Nunc)™)에 첨가하였다. PBS (pH 7.0) 내의 샘플을 1 mg/ml에서 분석하였다. 참조 샘플은 400 ㎕의 분석물과 동일한 완충제, 대체로 PBS를 함유하였다. 열 변성과 연관된 열 교환은 20℃ 내지 100℃에서 3.3℃/min의 가열 속도로 측정하였다. 겉보기 용융 온도 (Tm)는 50%의 분석물이 비폴딩되는 열 전이 중간지점에 대응하였다.
(
ix
) 혈청 안정성 연구
정제된 단백질을 0.3 mg/ml의 최종 농도가 되도록 35 ㎕ 래트 혈청 또는 마우스 혈청 (젠 텍스(Gene Tex))에 용해시켰다. 샘플을 37℃에서 플레이트 인큐베이터 내에서 인큐베이션하였다. 4 ㎕의 각각의 샘플을 상이한 시점에서 채취하고, 10 ㎕ 샘플 완충제 (4x, 누페이지, 인비트로젠) 및 26 ㎕ 물을 첨가하고, 샘플을 -20℃에서 동결시켰다. 12 ㎕의 각각의 샘플을 12% 비스-트리스-겔 (누페이지, 인비트로젠) 내로 로딩하고, 전기영동을 200V에서 35 min 동안 수행하였다. 단백질을 30V에서 1 h 동안 보레이트 완충제 (50 mM 보레이트, 50 mM 트리스) 내에서 PVDF-막 (인비트로젠)으로 이송하였다. 막을 TBST (10 mM 트리스-HCl, pH 7.5, 150 mM NaCl, 0.1% 트윈80) 내에서 잠깐 동안 세척한 다음, 2 h 동안 RT/진탕기에서 5% 분유를 함유하는 TBST 내에서 인큐베이션하였다. 막을 TBST 내에서 잠깐 동안 세척한 다음, POD-접합된 염소-항-인간 IgG, Fab 단편 특이적 (디아노바) (1:10,000 희석) 또는 항-His POD (로슈) (1:500 희석)를 함유하는 TBST 내에서 1 h 동안 RT/진탕기에서 인큐베이션하였다. 막을 5 min 동안 RT/진탕기에서 TBST 내에서 3회 세척하였다. 신호 검출은 BM 블루 POD 기질 (로슈) 또는 ECL/ECL 플러스 (지이 헬쓰케어)를 사용하여 이루어졌다.
(b) 결과
(i) 항-
LRP6
-
scFv
의 발현, 정제 및 특성 결정
모든 scFv는 이. 콜라이 W3110 내에서 성공적으로 발현되고, 친화도 크로마토그래피, 이어서 크기 배제 크로마토그래피에 의해 정제되었다. 예상된 크기 (MOR06475 (Gly4Ser)3/(Gly4Ser)4: 각각 26.74 및 27.06 kDa, MOR08168 (Gly4Ser)3/(Gly4Ser)4: 각각 26.5 및 26.85 kDa, MOR08545 (Gly4Ser)3/(Gly4Ser)4: 각각 25.99 및 26.31 kDa는 모든 정제된 샘플에 대해 환원 및 산화된 샘플을 사용하여 수행된 LC-MS 분석 및 SDS PAGE에 의해 확인되었다. >95%의 순도가 얻어졌다. 도 14는 SDS-PAGE 분석의 결과를 보여주고, 여기서 1.5 ㎍의 각각의 정제된 단백질은 10% 비스-트리스 겔 (누페이지, 인비트로젠) 내로 로딩하였다: M: 마커 씨 블루 플러스2 (See Blue Plus2) (인비트로젠); (1): MOR06475-VH-(Gly4Ser)4-VL; (2) MOR06475-VL- (Gly4Ser)4-VH; (3) MOR08168-VH-(Gly4Ser)4-VL; (4) MOR08168-VL-(Gly4Ser)4-VH; (5) MOR08545-VH-(Gly4Ser)4-VL; (6) MOR08545-VL-(Gly4Ser)4-VH; (7) MOR06475-VH-(Gly4Ser)3-VL; (8) MOR06475-VL-(Gly4Ser)3-VH; (9) MOR08168-VH-(Gly4Ser)3-VL; (10) MOR08168-VL-(Gly4Ser)3-VH; (11) MOR08545-VH-(Gly4Ser)3-VL; (12) MOR08545-VL-(Gly4Ser)3-VH.
열 안정성을 DSC에 의해 모든 ScFv에 대해 비교하였다. 용융 온도 (Tm)는 MOR06475 변이체에서 유의하게 더 높았다. 최고 Tm (64.7℃)은 MOR06475-VL-(Gly4Ser)4-VH에서 관찰되었고, 따라서 상기 분자는 생산된 다른 구축물보다 잠재적으로 더 안정한 것으로 생각된다.
MOR06475, MOR08168 및 MOR08545 scFv 및 Fab 구축물, 및 몇몇의 이중파라토프 포맷의 활성을 HEK293 STF 검정으로 평가하였다 (도 17 및 18). 전체적으로, 데이터는 STF 검정에서 프로펠러 1 IgG (MOR08168)가 프로펠러 3 리간드, 예컨대 Wnt3을 강화하면서 프로펠러 1 리간드, 예컨대 Wnt1을 억제함을 보여준다. 프로펠러 3 scFv6475는 프로펠러 1 리간드, 예컨대 Wnt1에 대한 활성을 갖지 않으면서 프로펠러 3 리간드, 예컨대 Wnt3을 억제한다. 또한, MOR08168/6475 이중파라토프 항체는 프로펠러 1 및 프로펠러 3 리간드 둘 모두에 대한 활성을 갖고, HEK293 Wnt STF 검정에 적용된 임의의 농도에서 강화 활성을 갖지 않는다.
(
ii
)
이중파라토프
항-
LRP6
IgG
-
scFv
의 발현, 정제 및 특성 결정
본 연구에서 생산된 이중파라토프 항-LRP6 Ig-scFv 포맷의 모식도가 도 15에 제시된다. 도 15A는 IgG의 C-말단에 부착된 scFv scFv를 나타내고; 15B는 Fc의 N-말단에 부착된 scFv scFv를 나타내고; 15C는 Fc의 C-말단에 부착된 scFv scFv를 나타내고; 15D는 Fc의 N- 및 C-말단에 부착된 scFv scFv를 나타낸다.
현재의 실험에서 이중파라토프 항체는 hIgG1 LALA CH3의 C-말단에 융합된 scFv를 갖는 이중파라토프 전장-IgG이다. 상기 특정 연구에서, scFv는 전장-IgG로부터 (GlyGlySer)2-링커에 의해 분리되었다. scFv는 (Gly4Ser)4 링커와 함께 VL-VH 배향으로 이루어진다.
이중파라토프 항-LRP6 IgG-scFv는 HEK293-6E 세포에서 일시적으로 발현시키고, pH 4.5 내지 2.5의 구배 용리를 사용하여 친화도 크로마토그래피에 의해 정제하였다. 197.4 kDa의 예상된 크기는 LC-MS 분석 및 SDS-PAGE에 의해 결정되었고, 순도는 97% 초과이었다. SEC MALS에 의해 결정된 응집은 5% 미만이다. 도 16은 정제된 이중파라토프 항-LRP IgG-scfV의 SDS-Page 분석이다. 샘플을 12% 비스-트리스 겔 (누페이지, 인비트로젠) 내로 로딩하였다. 마커: 인비트로젠 Mark12; (1): 비-환원; (2): 환원.
이중파라토프 항-LRP6 IgG scFv 및 모 항-LRP6 IgG는 비아코어에 의해 결정시에 각각 0.021 및 0.023 μM의 Kd로 pH 6.0에서 인간 FcRn에 결합하였다. 두 포맷은 pH 7.4에서 인간 FcRn에 대한 낮은 수준의 결합을 보였고, 따라서 상기 BpAb는 생체 내에서 표준 IgG로서 거동할 것으로 예상된다 (IgG1에 유사한 PK 특징) (도 19 참조).
이중파라토프 항-LRP6 IgG scFv는 336시간까지 시험할 때 37℃에서 마우스 및 래트 혈청 둘 모두에서 안정하고 (데이터 미제시), 384개의 정제된 세포외 또는 분비된 단백질을 함유하는 주문 제작 프로타겐 유니칩 상에서 평가할 때 >4%의 결합을 보이지 않았다 (데이터 미제시).
(c) 논의
최종 이중파라토프 항체의 최적화가 가능하도록 다양한 항-LRP6 scFv의 특성을 초기에 결정하였다. 두 배향으로 2개의 상이한 링커 길이를 갖는 ScFv를 이. 콜라이에서 발현시켰다. 항-LRP6 MOR06475, 8168 및 8545를 (Gly4Ser)3 및 (Gly4Ser)4 링커와 함께 VH-VL 및 VL-VH scFv로서 발현시켰다. 모든 MOR06475 및 MOR08168 변이체를 성공적으로 발현시키고, 응집체의 낮은 수준 (<5%)으로 정제하였다. 예상된 크기를 갖는 정확하게 프로세싱된 단백질을 얻었다. 열 안정성 데이터는 가장 안정한 scFv 포맷이 64.7℃의 Tm을 갖는 MOR06475-VL-(Gly4Ser)4-VH임을 보여주었다. 모든 시험된 MOR08168 scFv 포맷은 50-52℃의 Tm으로 유의하게 감소된 열 안정성을 보여주었다.
CH3 및 VL에 scFv를 갖는 이중파라토프 항-LRP6 IgG 및 변형된 변이체 (CH3에 C-말단 Lys (K)이 존재하지 않고, scFv의 VH에 AspPro (DP)의 AspAla (DA)로 및 AspPro (DP)의 ThrAla (TA)로의 치환이 존재하는)를 성공적으로 발현시켰고, 세포 배양액으로부터 응집체의 낮은 수준 (<5%)으로 정제하였다. 약 197-198 kDa의 예상된 크기가 결정되었다. VL에 구축물 항-LRP6 MOR08168 hIgG1LALA 6475scFv 및 항-LRP6 MOR08168hIgG1 LALA 6475scFv 및 돌연변이된 구축물 (CH3에서 C-말단 Lys (K)의 결실 및 AspPro (DP)의 AspAla (DA)로 및 AspAla (DP)의 ThrAla (TA)로의 치환)는 VL-VH 배향을 갖는 scFv로 이루어졌고, (Gly4Ser)4 링커에 의해 분리되었다. (GlyGlySer)2 링커를 사용하여 scFv를 hIgG1 LALA의 CH3 도메인에 및 h람다의 VL에 각각 부착시켰다. 그러나, 앞에서 논의된 바와 같이, scFv는 또한 다른 링커에 의해 분리된 VH-VL로 이루어질 수 있고, 또한 scFv는 CL의 C-말단 및 VH의 N-말단을 포함하는 IgG 내의 다른 위치에 다른 링커를 사용하여 부착될 수도 있다. MOR08168scFv를 갖는 구축물은 VH-VL 배향의 scFv로 이루어지고, (Gly4Ser)3 및 (Gly4Ser)4 링커에 의해 분리되었다. (GlyGlySer)2 링커는 scFv를 hIgG1 LALA의 CH3 도메인에 부착시키기 위해 사용되었다.
이중파라토프 항-LRP6 MOR08168 hIgG1LALA 6475scFv는 혈청에서 안정하였다 (336시간까지 시험될 때). 이중파라토프는 pH 6.0에서 인간 FcRn에 예상한 바와 같이 결합하였고, 낮은 수준 결합이 pH 7.4에서 관찰되었다. 모 항체는 유사한 동역학으로 결합하였다.
실시예
13:
이중파라토프
항체 항-
LRP6
MOR08168hIgG1LALA
6475
scfv
의 생체내 평가
생체 내에서 Wnt3 클래스 Wnt 신호 전달을 억제하는 이중파라토프 항체 항-LRP6 MOR08168hIgG1LALA 6475 scfv의 능력은 Wnt3A 분비 L 세포를 동시-이식한 PA1-STF 리포터 세포로 이루어지는 동시-이식편 시스템에서 시험하였다. 암컷 누드 마우스에게 10x10e6 PA1-STF 세포 및 0.5x10e6 L-Wnt3A 세포를 피하 이식하고, 5개의 군으로 무작위로 분류하였다. 24시간 후에, 마우스에게 단일 정맥내 용량의 비히클, MOR08168 LRP6-프로펠러 1 Ab (10 mg/kg), MOR06475 LRP6-프로펠러 3 Ab (10 mg/kg), 또는 이중파라토프 항체 항-LRP6 MOR08168hIgG1LALA 6475 scfv (1 mg/kg, 3 mg/kg, 또는 10 mg/kg)를 투여하고, 6시간, 24시간, 48시간, 72시간 및 168시간 후에 제노겐에 의해 영상화하였다. 이중파라토프 항체 항-LRP6 MOR08168hIgG1LALA 6475 scfv는 Wnt3A 유도 신호 전달의 용량-관련 억제를 보이고, 여기서 1 mg/kg이 24시간에서 최대 억제를 보이고, 48시간에 기준선으로 회복되었고, 3 mg/kg 및 10 mg/kg은 적어도 72시간 동안 지속적인 억제를 보였다. 10 mg/kg으로 투여된 MOR06475 LRP6-프로펠러 3 Ab는 적어도 72시간 동안 Wnt3a 유도 신호 전달을 억제할 수 있는 반면에, 10 mg/kg으로 투여된 MOR08168 LRP6-프로펠러 1 Ab는 Wnt3a 유도 신호 전달을 증가시켰다 (도 20).
MMTV-Wnt1 종양에서 Wnt 신호 전달에 대한 이중파라토프 항체 항-LRP6 MOR08168hIgG1LALA 6475 scfv의 효과를 MOR08168 LRP6-프로펠러 1 Ab과 비교하여 측정하기 위해, MMTV-Wnt1 종양을 이식한 마우스에게 단일 용량의 5 mg/kg의 이중파라토프 항체 항-LRP6 MOR08168hIgG1LALA 6475 scfv 또는 단일 용량의 5 mg/kg의 MOR08168 LRP6-프로펠러 1 항체를 i.v. 투여하였다. 이중파라토프 항체 및 프로펠러 1 항체의 혈청 농도 및 β-카테닌 표적 유전자 액신2의 mRNA 발현을 2주 기간에 걸쳐 분석하였다. 이중파라토프 항체의 말기 β-상 반감기는 약 48시간인 반면, LRP6 항체는 약 72시간이었다. 이중파라토프 항체 또는 프로펠러 1 항체를 투여한 마우스로부터 얻어진 종양에서 액신2 mRNA 발현의 유의한 감소가 관찰되었고, 액신2 발현은 점차 회복하여, 이중파라토프 항체 항-LRP6 MOR08168hIgG1LALA 6475 scfv와 MOR08168 LRP6-프로펠러 1 항체 사이에서 유의한 차이가 관찰되지 않았다 (도 21).
이중파라토프 항체 항-LRP6 MOR08168hIgG1LALA 6475 scfv의 항-종양 활성을 MMTV-Wnt1 동종이식편 모델에서 평가하였다. MMTV-Wnt1 종양 단편을 암컷 누드 마우스에 피하 (s.c.) 이식하였다. 이식 7일 후에, MMTV-Wnt1 종양을 보유한 마우스 (n=8, 평균 137 mm3; 범위: 81-272 mm3)를 비히클 IgG, MOR08168 LRP6-프로펠러 1 Ab (3 mg/kg, i.v., qw), 또는 이중파라토프 항체 항-LRP6 MOR08168hIgG1LALA 6475 scfv (3 mg/kg, i.v., 2qw)로 처리하고, 종양을 매주 2회 캘리퍼스로 측정하였다. MOR08168 LRP6-프로펠러 1 Ab 및 이중파라토프 항체 항-LRP6 MOR08168hIgG1LALA 6475 scfv 항체는 모두 종양 퇴행을 유도하였다 (각각 -93%, p<0.05 및 -91%, p<0.05) (도 22 참조). 이중파라토프 항체 항-LRP6 MOR08168hIgG1LALA 6475 scfv의 용량 의존성을 평가하고, 도 23에 도시한다.
β-카테닌 siRNA 또는 우성-음성 TCF-4에 의한 결장직장 암 세포에서 Wnt 신호 전달의 억제는 신속한 세포 주기 정지를 일으키고, 창자 분화 프로그램을 유도한다 ([van der Wetering et al. (2002), Cell, 111, 241-250]; [van der Wetering et al. (2003), EMBO Reports, 4, 609-615]). 뮤린 MMTV-Wnt1 유방 종양에서 길항 LRP6 항체에 의한 Wnt 신호 전달의 억제가 유사한 결과를 갖는지 결정하기 위해, 분비 분화를 우유의 주요 성분인 지질에 대한 오일 레드 O 염색에 의해 검사하였다. MMTV-Wnt1 종양 보유 마우스를 단일 용량의 PBS (대조군) 또는 5 mg/kg 항-LRP6 MOR08168hIgG1LALA 6475 scfv로 처리하고, 처리 24 h, 72 h 또는 5일 후에 동결된 뮤린 종양 동종이식편의 절편을 5 ㎛ 두께로 절단하였다. 슬라이드를 30-60분 동안 실온에서 공기 건조시킨 후, 빙냉 10% 포르말린 내에서 5-10분 동안 고정시켰다. 슬라이드를 즉시 dH2O 내에서 3회 세정하였다. 오일 레드 O 염색을 오일 레드 O 염색 키트 (폴리 사이언티픽 알앤디 (Poly Scientific R&D), Cat # k043)를 사용하여 수행하였다. 슬라이드를 스캔스코프 (ScanScope) CS/GL 스캐너 (아페리오 테크놀로지스 (Aperio Technologies))로 스캐닝하고, 조직 절편을 양성-화소 카운트와 함께 IHC 색상 데콘벌루션 (deconvolution) 알고리즘을 사용하여 이미지스코프 (ImageScope) v10.2.1.2315 소프트웨어 (아페리오 테크놀로지스)로 분석하였다. 오일 레드 O 염색의 대표적인 영상을 도 24a에, 정량을 도 24b에 제시한다. 그래프는 평균±SEM 값을 나타낸다. 72시간 군에서 n=4, 24시간 군에서 n=3, 5일 군에서 n=2, 및 PBS (대조군)에 대해 n=1이고, 실험의 시간 과정 동안 오일 레드 O 염색의 증가를 보인다. 함께 살펴보면, 상기 결과는 유방 종양 세포에서 Wnt 신호 전달의 억제는 세포 주기 정지 및 분비 분화 프로그램의 유도를 일으킬 수 있음을 제안한다.
이중파라토프 항체 항-LRP6 MOR08168hIgG1LALA 6475 scfv의 항-종양 활성을 MDA-MB-231 유방 이종이식편 모델에서 추가로 평가하였다. 50% 마트리겔 내의 5x10e6 MDA-MB-231 세포를 암컷 누드 마우스 내로 피하 (s.c.) 이식하였다. 이식 31일 후에, MDA-MB-231 종양을 보유한 마우스 (n=7, 평균 165 mm3; 범위 99-238 mm3)를 비히클, 또는 이중파라토프 항체 항-LRP6 MOR08168hIgG1LALA 6475 scfv (3 mg/kg, i.v., qw)로 처리하고, 종양을 매주 2회 캘리퍼스로 측정하였다. 매주 3 mg/kg으로 투여된 이중파라토프 항체 항-LRP6 MOR08168hIgG1LALA 6475 scfv 항체는 종양 성장을 유의하게 지연시켰다 (T/C=23%, p<0.05) (도 25 참조).
실시예
14:
MMTV
-
Wnt1
모델에서 추가의
이중파라토프
항체의 생체내 평가
실시예 10에 기재된 바와 같이, 프로펠러 3 항체 MOR06475 및 프로펠러 1 MOR08168 scfv 도메인으로 이루어진 추가의 항-LRP6 역 이중파라토프 항체를 생성하였다. MOR08168hIgG1LALA 6475 scfv에 비해, 생체 내에서 Wnt1 신호 전달을 억제하는 상기 이중파라토프 항체 (MOR06475hIgG1 LALA_8168scfv_(VH-3-VL) 및 MOR06475hIgG1 LALA_8168scfv_(VH-4-VL))의 능력을 MMTV-Wnt1 모델에서 결정하였다. MMTV-Wnt1 종양을 이식한 마우스에게 단일 용량의 5 mg/kg의 상기 기재된 각각의 항체를 i.v. 투여하였다. 각각의 항체 (MOR08168hIgG1 LALA 6475 scfv, MOR06475hIgG1 LALA_8168scfV_(VH-3-VL), 및 MOR06475hIgG1 LALA_8168scfv_(VH-4-VL))의 혈청 농도, 및 β-카테닌 표적 유전자 액신2의 mRNA 발현을 5일 기간에 걸쳐 분석하였다 (평가된 시점은 0, 2, 7, 24, 72 및 120 h이었다). 두 역 이중파라토프 항체는 모두 MOR08168hIgG1LALA 6475 scfv와 동일한 최대 정도로 액신2 mRNA 발현의 유의한 감소를 보였다. 그러나, 액신2 mRNA 발현 감소의 지속기간은 MOR08168hIgG1 LALA 6475 scfv를 사용하여 관찰된 것보다 더 짧았고, 신호는 두 역 이중파라토프 분자에 대해 24 h 시점에서 기준선으로 회복하였다. 이것은 상기 분자의 노출 감소와 일치하고, 혈청 수준은 MOR08168hIgG1 LALA 6475 scfv에 대한 120 h에 비해 24 h에서 5 ㎍/ml 미만으로 감소하였다.
실시예
15: 재조합
LRP6
PD1
/2 및
PD
3/4에 대한
이중파라토프
항체의 결합 친화도
LRP6의 프로펠러 도메인 1-2 (PD1/2, 기탁 번호 NP002327의 아미노산 잔기 19 내지 629) 및 LRP6의 프로펠러 도메인 3-4 (PD3/4, 기탁 번호 NP002327의 아미노산 631-1246)에 대한 항-LRP6 MOR08168hIgG1 LALA 6475 scfv, MOR08168, 및 MOR6475의 결합 친화도를 비아코어 T-100 (지이 헬쓰케어)을 사용하여 표면 플라스몬 공명 (SPR)을 통해 평가하였다. 친화도 결정을 위해, 항-인간 IgG Fcγ 특이적 항체 (#109-005-098, 잭슨 이뮤놀로지 (Jackson Immunology))를 10 mM 아세트산나트륨 (pH 5.0) 완충제 내에 희석한 후, 표준 아민 커플링 화학을 사용하여 모든 4-유동 세포에 대해 ~2000 RU의 밀도로 CM4 칩 (지이 헬쓰케어, BR-1005-34) 상에 고정하였다. 카르복시메틸 덱스트란 표면을 1:1 비율의 0.4 M EDC (1-에틸-3-[3-디메틸아미노프로필] 카르보디이미드 염산염) 및 0.1 M NHS (N-히드록시숙신이미드)를 7 min 주사하여 활성화하였다. 이어서, 과량의 반응성 에스테르를 1M 에탄올아민으로 차단하였다. 항-LRP6 MOR08168hIgG1LALA 6475 scfv를 HBS-EP 완충제 (지이 헬쓰케어) 내에 10 ㎍/ml로 제조하고, 유량 10 ㎕/min에서 ~70 RU의 밀도로 CM4 칩의 별개의 유동 세포 상에 포획하였다. PD1/2 및 PD3/4를 50 nM에서 출발하여 2배 농도 계열로 제조하고, 30 ㎕/min로 1 min 동안 주사하였다. 이것은 항-LRP6 MOR08168hIgG1LALA 6475 scfv-포획된 표면 및 포획된 리간드가 없는 대조군 표면 상에 대한 40 min 해리기를 허용하였다. 이어서, 표면을 10 mM 글리신 (pH 2.2)으로 2회 재생하였다. PD1/2 및 PD3/4에 대한 항-LRP6 MOR08168hIgG1LALA 6475 scfv의 이중 결합 분석을 위해, 항-LRP6 MOR08168hIgG1LALA 6475 scfv를 항-인간 IgG Fcγ 특이적 항체가 고정된 CM4 칩 상에 포획하였다. 이어서, 100 nM의 포화 농도의 PD3/4를 30 ㎕/min의 유량으로 30 min 동안 표면 상에 유동시켰다. 포화 농도의 100 nM PD1/2를 PD3/4 다음에 즉시 주사하였다. 해리 상수 (KD), 회합 (kon), 및 해리 (koff) 속도는 비아이밸류에이션(BIAevaluation) 소프트웨어 (지이 헬쓰케어)를 사용하여 기준선 공제 보정 결합 곡선으로부터 계산하였다.
PD1/2 및 PD3/4에 대한 항-LRP6 MOR08168hIgG1 LALA 6475 scfv (이중파라토프 항체) 결합을 MOR08168 (프로펠러 1 항체) 및 MOR6475 (프로펠러 3 항체)와 비교하였다. 도 26a는 상응하는 LRP6 수용체 도메인 PD1/2 및 PD3/4에 대한 분자의 친화도를 보여준다. PD1/2 및 PD3/4에 대한 항-LRP6 MOR08168hIgG1LALA 6475 scfv의 결정된 KD는 PD1/2에 대한 MOR08168 및 PD3/4에 대한 MOR6475의 것과 각각 유사하였다. 도 26ba 및 26bb는 각각의 단백질에 결합하는 항-LRP6 MOR08168hIgG1LALA 6475 scfv의 회합 및 해리상을 보여준다. PD1/2에 대한 항-LRP6 MOR08168hIgG1LALA 6475 scfv의 결합에 대한 오프-레이트는 PD3/4에 대한 것보다 더 느리다. PD1/2 및 PD3/4가 순차적으로 주사된 추가의 연구는 예상되는 바와 같이, 항-LRP6 MOR08168hIgG1LALA 6475 scfv가 두 프로펠러 도메인 구축물 모두에 결합할 수 있음을 나타내었다 (도 26c).
실시예
16:
scFv08168
및
scFv06475
의 열 안정성을 개선하기 위한
scFv
돌연변이
본 실시예는 프로펠러 1 및 프로펠러 3 항체의 scFv에서 이루어진 돌연변이 및 열 안정성에 의해 결정될 때 scFv의 안정성에 대한 개개의 돌연변이 및 돌연변이의 조합의 효과를 설명한다. scFv의 안정성 개선은 돌연변이된 scFv를 포함하는 항체 구축물의 전체 안정성으로 해석될 수 있다.
물질 및 방법
구축물
IgG 기반 이중파라토프 분자를 위해, scFv06475를 GlyGlySer 링커를 통해 MOR08168 IgG1의 C-말단에 융합시켜 도 27에서 "901"로 지정된 이중파라토프 항체 (MOR08168IgG1LALA 6475 scfv)를 만들거나, 또는 scFv08168을 GGS 링커를 통해 MOR06475 IgG1의 C-말단에 융합시켜 도 27에서 "902"로 지정된 이중파라토프 항체를 만들었다. 상세한 정보에 대해서는, 상기 특허 출원의 다른 부분을 참조한다.
scFv06475
및
scFv08168
에 대한 집중 (
focused
) 라이브러리의 합리적인 설계
scFv06475 및 scFv08168을 안정화하기 위한 점 돌연변이의 선택을 위해 다음과 같은 2개의 방법을 사용하였다: 서열 컨센서스 분석 및 MOE (Molecular Operating Environment)의 상동성 모델링을 사용한 구조 기반 돌연변이 설계.
서열 컨센서스 분석을 위해, scFv06475의 VH 및 VL 도메인의 아미노산 서열,및 scFv08168의 VH 및 VL 도메인의 아미노산 서열을 NCBI의 비-풍부 (non-redundant) 단백질 서열 데이터베이스에 대해 BLAST를 실행하였다. 각각의 BLAST 실행 후에, 질의 서열 및 상위 250개의 상동성 서열을 clustalW 프로그램에 의해 정렬하였다. 자체 (in-house) 컴퓨터 프로그램을 사용하여 정렬된 서열 중의 모든 잔기 위치에서 가장 통상적인 아미노산을 계수하였다. 질의 서열 내의 아미노산이 정렬된 서열 풀에서 가장 통상적인 아미노산과 상이한 각각의 위치에서, 돌연변이는 그의 야생형 아미노산으로부터의 잔기를 가장 통상적인 아미노산으로 돌연변이시키도록 설계되었다.
scFv06475의 상동성 모델 및 scFv08168의 상동성 모델은 MOE에서 구축되었다. 서열을 먼저 MOE의 "서열 편집기 (editor)" 모듈로 판독하였다. 상동성 서열을 갖는 존재하는 X-선 구조를 MOE의 "항체 모델러" 모듈에서 검색하였다. X-선 구조 3L5X 및 1W72가 각각 scFv06475 및 scFv08168에 대한 상동성 모델을 구축하기 위한 적합한 주형으로서 MOE에 의해 확인되었다. 이어서, 상동성 모델은 에너지를 최소화하기 위해 CHARMM27 역장 (force field)을 사용하여 MOE에 의해 구축하였다.
이어서, MOE에 의해 생성된 모델을 NAMD에서 에너지 최소화 및 MD 시뮬레이션의 5개 단계에 적용하였다. 전체 모델은 제1 내지 제3 단계에서 5000 단계 동안 에너지 최소화되고, 30 kcal/mol/A2의 리스트레인트 (restraint)가 상이한 세트의 원자에 적용되었다. 제1 단계에서, 리스트레인트는 CDR 내의 측쇄 원자를 제외한 모든 원자에 적용되었다. 제2 단계에서, 리스트레인트는 CDR 내의 잔기를 제외한 모든 원자에 적용되었다. 제3 단계에서, 리스트레인트는 주쇄 원자에만 적용되었다. 이어서, 모델을 제4 단계에서 50K에서 100 ps에 대해 진공에서 시뮬레이션하였고, 여기서 30 kcal/mol/A2의 리스트레인트가 주쇄 원자에 적용되었다. 마지막 제5 단계에서, 모델은 5000개 단계에서 에너지 최소화되었고, 30 kcal/mol/A2의 리스트레인트가 주쇄 원자에 적용되었다. 상기 5개의 최소화 및 MD 시뮬레이션 단계 후의 모델은 구조 기반 돌연변이 설계를 위한 상동성 모델로서 간주되었다.
돌연변이체 모델을 야생형 상동성 모델에 대해 구축하였다. VMD의 "psfgen" 모듈의 "돌연변이시켜라 (mutate)" 명령을 사용하여 잔기를 설계된 아미노산으로 돌연변이시켰다 (William Humphrey et al. (1996) J. Molecular Graphics, 14:33-38). 이어서, 돌연변이된 모델을 에너지 최소화시키고, 6개의 단계에서 시뮬레이션시켰다. 에너지 최소화의 5000 단계를 제1 내지 제4 단계에서 수행하였다. 제1 단계에서 30 kcal/mol/A2의 리스트레인트를 돌연변이된 잔기의 측쇄를 제외하고 모든 원자에 대해 적용하였다. 제2 단계에서 동일한 강도의 리스트레인트를 돌연변이된 잔기를 제외하고 모든 원자에 대해 적용하였다. 돌연변이된 잔기의 5Å 내의 잔기의 측쇄 원자에 대한 리스트레인트는 제3 단계에서 제거되었다. 또한, 돌연변이된 잔기의 5Å 내의 잔기의 주쇄 원자에 대한 리스트레인트는 제4 단계에서 방출되었다. 제4 단계에서 적용된 것과 동일한 리스트레인트로, 돌연변이된 모델을 50K에서 100 ps에 대해 진공에서 시뮬레이션하였다. 이어서, 시뮬레이션 궤적의 마지막 스냅샷은 다시 제4 단계에서와 동일한 리스트레인트로 5000개 단계에서 에너지 최소화되었다. 상기 최소화된 모델은 돌연변이체의 상동성 모델로서 간주되었다.
이.
콜라이
시스템에서 플레이트 기반-라이브러리 제작, 발현 및 정제
고처리량 돌연변이 유발을 퀵체인지 XL 부위-지정 돌연변이 유발 키트 (스트라타젠)를 사용하여 수행하였다. 프라이머는 프라이머 설계 소프트웨어 뮤타프라이머 (Mutaprimer)에 따라 설계되었고, IDT로부터 표준 농도로 96웰 포맷으로 주문되었다. 돌연변이체 표준 합성 반응 부피는 50 ㎕로부터 25 ㎕로 축소하였다. 반응은 96웰 PCR 플레이트에서 수행하였다. 순환 후에, 0.5 ㎕ DpnI 효소를 각각의 증폭 반응액에 첨가하고, 37℃에서 2 h 동안 인큐베이션하여 모 dsDNA를 소화시켰다. 형질전환은 96웰 PCR 플레이트에서 2 ㎕의 DpnI 소화시킨 돌연변이 유발 반응액을 20 ㎕ 아셀라 (Acella) 화학 전능성 세포에 첨가함으로써 수행하였다.
발현 및 정제를 위해 각각의 형질전환 플레이트로부터 3개의 개별적인 콜로니를 선발하였다. 발현은 자가유도 배지를 사용하여 2개의 96웰 딥-웰 플레이트에서 수행하였다. 박테리아 배양액의 분취액을 글리세롤 원액으로서 보관하고, 서열결정 분석을 위해 보냈다. 각각의 개개의 콜로니에 대한 2개의 플레이트로부터 조합된 박테리아 펠릿을 용해시키고, MagneHis 단백질 정제 시스템 (프로메가)으로 정제하였다. 킹피셔 (KingFisher) 기기를 고처리량 정제로 설정하였다. 1 M NaCl을 단백질 순도를 개선하기 위해 용해 및 세척 완충제에 첨가하였다. 단백질을 PBS 내의 100 ㎕의 300 mM 이미다졸로 용리하였다. 단백질 양을 시차 주사 형광 (Differential Scanning Fluorimetry; DSF)을 위한 단백질의 최적량을 결정하기 위해 쿠마지 플러스 (Coomasie plus) (써모 (Thermo))로 짧게 조사하였다.
DSF
및 시차 주사 열량분석 (
DSC
)에 의한
열안정성
돌연변이의 스크리닝
각각의 샘플에 대해 정제된 단백질 양에 따라, 대체로 10 내지 20 ㎕의 용리액이 DSF 분석을 위해 사용되었다. 구체적으로, 10-20 ㎕의 샘플을 PBS 내의 25 ㎕의 총 부피에서 1:1000의 최종 희석율의 시프로 오렌지 (Sypro Orange) (인비트로젠)와 혼합하였다. 샘플을 바이오라드 (BioRad) CFX1000 (2 min 동안 25℃, 이어서 30초 동안 25에서 95℃로 0.5℃씩 증가시킴)에서 시험하였다. 적중치는 야생형 scFv보다 2℃ 더 높은 Tm으로서 규정되었다. scFv 결합 활성은 원-포인트 ELISA에 의해 결정하였다.
포유동물 세포에서 단백질 생산
His 태그와 함께 scFv06475 또는 scFv08168을 갖는 포유동물 구축물 pRS5a 내로 돌연변이를 도입하였다. 구축물을 50 ml의 293T 현탁액 세포에서 일시적으로 발현시켰다. 간단히 설명하면, PEI를 최적 형질감염 효율을 위해 DNA 50 ㎍과 1:3으로 혼합하였다. 1.4 e6/ml의 세포를 형질감염을 위해 사용하였다. 형질감염된 세포를 250 ml의 여과지 플라스크에서 80 rpm으로 진탕하면서 CO2 챔버에서 6일 인큐베이션한 후에 수집하였다. 상청액을 최적 단백질 회수를 위해 약 1 ml로 농축시켰다. 단백질을 제조자의 지시에 따라 MagneHis 키트에 의해 손으로 정제하였다. 정제된 단백질을 완충제를 변경하면서 PBS 내에서 밤새 투석하였다. 투석 전 및 후의 단백질 샘플을 DSF 분석을 위해 사용하였다.
scFv
에 대한 친화도 측정
LRP6 단백질에 대한 scFv의 결합 EC50을 ELISA에 의해 측정하였다. 맥시소르프 플레이트를 LRP6-Fc (알앤디 시스템즈 (R&D Systems), 카탈로그 No: 1505-LR)로 3 ㎍/ml에서 4℃에서 밤새 코팅하였다. 플레이트를 50 ㎕의 2% BSA로 1시간 동안 차단하고, 세척 용액으로 5회 세척하였다. 샘플은 이에 따라 1% BSA로 희석되었다. 플레이트를 RT에서 1 h 동안 인큐베이션하고, 3회 세척하였다. 검출은 1% BSA 내에 1:2000 희석액으로 50 ㎕ 펜타His-HRP (퀴아젠 Mat. No. 1014992)를 첨가하여 수행하고, RT에서 1 h 동안 인큐베이션하고, 3회 세척하였다. 50 ㎕의 기질 시약 A + B (알앤디 시스템즈)를 첨가한 후, 색상에 따라 5-20 min 동안 인큐베이션하였다. 25 ㎕의 중지 용액을 첨가하여 반응을 중지시킨 후, 플레이트를 450 nm에서 판독하였다.
동역학 실험을 바이오라드의 프로테온 XPR36 바이오센서를 사용하여 수행하였다. 모든 실험을 이동 완충제로서 PBST (0.05% 트윈-20이 존재하는 인산염 완충 염수)를 사용하여 실온에서 수행하였다. GLM 칩 상의 6개의 모든 수직 채널은 EDC (400 mM) 및 sNHS (100 mM)의 새로 제조한 혼합물을 사용하여 30 ㎕/min의 유량에서 5 min 동안 활성화시켰다. 항-His 마우스 IgG1 (알앤디 시스템즈, 카탈로그 No: MAB050)을 10 mM 아세트산나트륨 (pH 5.0)에 20 ㎍/ml로 희석하고, 30 ㎕/min의 유량에서 별개의 수직 채널을 따라 5 min 동안 칩에 커플링시켰다. 이어서, 1 M 에탄올아민을 30 ㎕/min에서 5 min 동안 주사하여 미반응 sNHS 기를 실활시켰다. 이어서, 2 ㎍/ml MOR08168 scFv 야생형 또는 2 ㎍/ml MOR08168 D1 돌연변이체를 100 ㎕/min에서 15초 동안 상이한 수직 채널에 고정시키고, 수평 방향으로 30 ㎕/min의 이동 완충제의 1 min 주사를 2회 실시하였다. 제6 수직 채널을 채널 참조물로 사용하였고, 어떠한 리간드도 상기 채널에 고정되지 않았다. 360 KD 동종이량체 항원 LRP6-Fc (알앤디 시스템즈, 카탈로그 No: 1505-LR)의 희석 계열을 300, 100, 33, 11, 및 3.7 nM의 최종 농도로 제조하고, 각각의 수평 채널을 따라 30 ㎕/min으로 주사하였다. 결합을 5 min 동안 모니터링하고, 해리를 20 min 동안 모니터링하였다. 완충제를 실시간 기준선 변화 교정 (drifting correction)을 위한 행 참조물 (row reference)로서 기능하기 위해 제6 채널에 주사하였다. 칩 표면은 수평 방향으로 18초 동안 100 ㎕/min으로 0.85% 인산을 적용하고 수직 방향으로 동일한 이동 조건에 의해 재생하였다. 프로테온의 동역학 분석을 프로테온 매니저 (Manager) v.2.1.1에서 수행하였다. 실시간 기준선 변화를 교정하기 위해 각각의 상호작용 스팟 데이터에서 채널 참조물, 이어서 행 참조물을 뺐다. 처리된 데이터는 이가 분석물 모델에 전체적으로 피팅되었다.
결과
이.
콜라이에서
플레이트-기반 돌연변이 유발, 발현 및 정제는 집중 라이브러리로부터 보다
효율적인 스크리닝을 허용한다.
하류 분석을 용이하게 하기 위해 고수율을 달성하기 위해, 발현에 대한 리더 (leader) 서열의 효과를 시험하였다. pelB와 함께 리더는 도 28A에 제시된 바와 같이 시험한 7개의 리더 중에서 최고량의 정제된 단백질을 생성함이 밝혀졌다. BL21 (DE3), XL-1 블루 및 W3110을 포함하는 몇몇 박테리아 균주를 IPTG 유도에 의한 발현에 대해 시험하였다. BL21 (DE3)에서 scFv06475의 발현 수준은 도 28B에 제시된 바와 같이 XL-1 블루에서보다 더 높았고, W3110에서보다는 약간 더 높았다. 돌연변이 유발 클로닝, 형질전환 및 발현을 용이하게 하기 위해, 아셀라 (BL21의 유도체)를, 클로닝 및 발현이 고효율로 동일한 균주에서 수행될 수 있기 때문에 모든 후속 실험에서 사용하였다. 단백질을 MagneHis 키트를 사용하여 킹피셔에 의해 세포 용해물로부터 정제하였다. scFv08168에 대한 추정 수율은 딥 웰 배양 플레이트로부터 조합된 웰의 샘플당 약 10 ㎍이었고, 그 중 2 ㎍을 DSF 열안정성 분석을 위해 사용하였다. 플레이트 기반 HTP 스크리닝은 프라이머로부터 개선된 열안정성을 갖는 적중치까지 시간을 약 1주로 유의하게 단축시켰다.
집중 라이브러리로부터 단일 점 돌연변이에 의한
scFv
열안정성
개선
개선된 열안정성에 대한 적중치는 지속적으로 야생형보다 적어도 1℃ 더 높은 Tm의 개선으로서 규정되었다. scFv06475에 대해 총 51개의 서열 확인된 변이체로부터 9개의 적중치 및 scFv08168에 대해 총 83개의 서열 확인된 변이체로부터 15개의 적중치가 존재하였다. 선택된 적중치를 scFv06475에 대해 도 28에 및 scFv08168에 대해 도 29에 제시하였다.
scFv06475 및 scFv08168을 안정화하기 위해 점 돌연변이의 선택을 위해 다음과 같은 2개의 접근법이 사용되었다: 서열 컨센서스 분석 및 구조 기반 돌연변이 설계. scFv06475의 VH 도메인에 상동성인 상위 250개의 서열 중에서, VH: 34 위치는 Met (45%) 또는 Val (48%)에 의해 통해 점유되었다 (모든 넘버링 시스템은 카바트 시스템에 따른 것이다). 또한, 상기 위치는 scFv06475의 야생형 서열에서 Gly 잔기이었다. 2개의 돌연변이체, 즉 VH:G34M 및 VH:G34V는 야생형 아미노산을 상기 위치에서 보다 인기있는 아미노산으로 돌연변이시키도록 설계되었다. 도 28에 나열된 바와 같이, VH:G34V 돌연변이체는 2개의 상이한 프로토콜에 의해 발현되고 정제될 때 야생형 scFv06475보다 더 높은 안정성을 지속적으로 보였다. VH:G34M 돌연변이체는 아마도 PCR 절차에서 오류에 의해 발생하는 틀린 서열에 의해 박테리아에서 잘 발현되지 않았다.
동일한 서열 컨센서스 분석에 기초하여, VH:I34M, VH:G50S, VH:W52aG 및 VH:H58Y의 돌연변이는 그의 상위 250개의 상동성 서열에서 scFv08168 내의 잔기를 컨센서스 아미노산으로 돌연변이시키도록 설계되었다. 도 30에 제시된 바와 같이, 상기 돌연변이는 scFv08168의 Tm을 각각 7.5℃, 3.0℃, 7.0℃, 및 3.5℃ 개선하였다.
구조 기반 접근법에서, scFv06475의 상동성 모델 및 scFv08168의 상동성 모델을 먼저 MOE로 구축한 후, NAMD로 에너지 최소화하였다. 이어서, 상기 모델을 국소 상호작용을 향상시키는 잠재적인 돌연변이에 대해 가시적으로 조사하였다. scFv06475 및 scFv08168의 다양한 잔기에 대한 돌연변이를 단백질 안정성에 대한 5가지 생물물리적 이해를 기초로 하여 설계하였다.
제1 접근법은 충전을 개선하기 위해 단백질 코어 내의 측쇄의 크기를 증가시키는 것이다. 단백질 코어에 대면하는 측쇄를 갖는 일부의 소수성 잔기는 상기 잔기 주위의 충전을 개선하기 위해 보다 큰 측쇄로 돌연변이되었다. 스크리닝 후에, 상기 방법에 의해 설계된 3개의 돌연변이는 scFv08168의 안정성을 개선하는 것으로 밝혀졌다. 도 30에 나열된 바와 같이, VH:I34F, VL:V47L 및 VL:G64V 돌연변이는 scFv08168의 용융 온도를 각각 4.0℃, 2.5℃, 및 2.0℃ 개선하였다.
제2 접근법은 파이-파이 적층 상호작용을 형성하도록 소수성 잔기를 방향족 잔기로 돌연변이시키는 것이었다. 도 33a에 제시된 바와 같이, 야생형 scFv06475 상동성 모델에서, VH:I37 잔기의 측쇄는 VH:W103 및 VL:F98의 2개의 방향족 잔기에 매우 근접하여 위치하였다. VH:I37의 임의의 비-수소 측쇄 원자와 VH:W103의 임의의 비-수소 측쇄 원자 사이의 가장 가까운 거리는 3.82Å이었다. VH:I37과 VL:F98 사이의 대응물 거리는 3.77Å이었다. 도 33b에 제시된 바와 같이, 2개의 수직의 파이-파이 적층 상호작용은 Phe 잔기가 VH:I37F 돌연변이를 통해 상기 국소 영역에 도입될 때 형성되었다: VH:F37과 VH:W103 사이의 하나, 및 VH:F37과 VL:F98 사이의 하나. 새로 형성된 파이-파이 적층 상호작용은 scFv6475의 용융 온도가 상기 돌연변이에 의해 61에서 64.5℃로 개선되었기 때문에 상기 국소화 영역에서 원래의 소수성 상호작용보다 더 강해야 한다 (도 28). VH:M95F 돌연변이는 또한 VH:W50 및 VH:F100과 파이-파이 적층 상호작용을 형성함으로써 scFv06475의 안정성을 개선하였다 (도 29). Tm은 61에서 64.5℃로 개선되었다.
제3 접근법은 염 가교를 형성하기 위해 비-하전된 잔기를 하전된 잔기로 돌연변이시키는 것이었다. scFv6475의 VH:K43 잔기는 상동성 모델에서 이웃 잔기와 염 가교를 형성하지 않았다. VH:V85 측쇄는 scFv06475의 상동성 모델에서 VH:K43에 직접 대면하기 때문에 (도 33e), VH:K43과 염 가교를 형성하기 위해서 음 하전되도록 돌연변이되었다. 도 33f에 제시된 바와 같이, 돌연변이된 VH:E85 측쇄 비-수소 원자와VH:K43 측쇄 비-수소 원자 사이의 거리는 2.61Å 정도로 짧을 수 있고, 이것은 염 가교가 2개의 잔기 사이에서 확립될 수 있음을 제안한다. 도 29에 제시된 VH:V85E 돌연변이에 대한 scFv6475의 개선된 안정성은 실제로 상기 위치에서의 설계 근거를 지지하였다.
제4 접근법은 수소 결합을 확립하기 위해 소수성 잔기를 극성 잔기로 돌연변이시키는 것이었다. 도 33c에 예시된 바와 같이, 소수성 VH:V33 잔기는 scFv08168의 상동성 모델에서 극성 잔기 VH:N100a에 근접하였다. 극성 잔기가 VH:V33N 돌연변이를 통해 상기 영역에 삽입될 때, 추가의 수소 결합이 VH:N33과 VH:N100a 형성될 수 있다. 상동성 모델링은 VH:N33의 ND2 원자와 VH:N100a의 OD 원자 중의 하나 사이의 거리가 도 33d에 제시된 바와 같이 수소 결합의 범위 내인 2.80Å 정도로 짧을 수 있음을 제안하였다. 도 29에 제시된 바와 같이, 상기 VH:V33N 돌연변이는 scFv08168의 안정성을 2.0℃ 개선하였다. 마찬가지로, scFv06475에 대한 VL:D93N 돌연변이의 안정성 향상 (도 29 참조)은 VL:Q27 및 VL:Q90을 갖는 개선된 수소 결합의 기하학적 구조에 의한 것일 수 있다.
scFv08168의 상동성 모델에서 VL:T78 및 VH S49의 2개의 극성 잔기 둘 모두가 순수한 소수성 측쇄에 의해 둘러싸인다는 놀라운 관찰을 기초로 하여, 제5 방법이 다른 소수성 환경 내의 2개의 극성 잔기를 비-극성 잔기로 돌연변이시키기 위해 이용되었다. VL:T78 및 VH:S49의 2개의 극성 잔기 둘 모두는 순수한 소수성 측쇄에 의해 둘러싸였다. 도 29에 나열된 바와 같이, VL:T78V 및 VH:S49A 돌연변이는 scFv08168의 용융 온도를 각각 2.5℃ 및 5.5℃ 증가시켰다.
원 포인트 ELISA를 결합 활성을 평가하기 위해 수행하였다. 특정 적중치는 LRP6을 향한 결합 활성의 감소 또는 상실 때문에 제외하였다. 예를 들어, scFv08168의 돌연변이 VH W052aG는 Tm을 7℃ 개선하였지만, 야생형 scFv08168에 비해 훨씬 감소된 결합 활성을 보였다. 따라서, 이것은 추가의 분석을 위해 선택하지 않았다.
용리 완충제 내의 300 mM 이미다졸의 존재는 scFv06475에서 관찰되는 바와 같이 때로 DSF에 의한 Tm의 전체적인 전환을 유발하였다. 그러나, 이 효과는 Tm의 순위에 영향을 주지 않았다. 또한, 도 28에 제시된 바와 같이 scFv06475에 대해 포유동물 세포에 비해 이. 콜라이에서 생산된 단백질에 차이가 존재하였다. 반대로, 이미다졸의 존재는 도 30 및 도 31에 제시된 바와 같이 scFv8168의 Tm에 대해 최소의 효과를 갖는다. 또한, Tm 값은 도 31에 제시된 바와 같이 scFv08168에 대해 이. 콜라이 또는 포유동물로부터 생산된 단백질에서 변하지 않은 상태로 유지되었다. Tm 전환의 존재 여부와 상관없이, Tm 순위는 동일하게 유지되었다.
포유동물 세포에서 생산된 단백질에서 열안정성의 효과를 확인하기 위해서, 상기 돌연변이를 포유동물 발현 벡터의 구축물 내로 도입하고, 293T 현탁액 세포에서 발현시켰다. 이들을 MagneHis 비드에 의해 정제하였다. Tm을 조사하고, scFv08168에 대해 도 31에 제시된 바와 같이 포유동물 세포에서 발현된 단백질의 개선된 열 안정성에 대해 적중치를 확인하였다. scFv08168에 대해 최고 열안정성 개선을 갖는 단일 점 돌연변이는 7.5℃의 개선을 보이는 VH:I34M이다.
열안정성을
추가로 개선하기 위한 돌연변이의 조합
열안정성을 추가로 개선하기 위해, scFv의 열안정성을 개선한 단일 돌연변이를 조합하여 scFv08168에 이중 돌연변이를 만들었다. 도 31에 제시된 바와 같이, 상가적 효과가 D1을 포함하는 대부분의 이중 돌연변이체에서 관찰되었고, 이것은 VH:I34M 및 VH:S49A의 2개의 돌연변이의 조합에 의해 12.5℃의 개선을 보인 반면에, 단일 돌연변이는 각각 Tm의 7.5 및 5.5℃ 증가를 유발하였다.
결합 및 기능적 활성에 대해 특성화된
열안정성
돌연변이체
친화도 분석 ELISA EC50을 scFv08168 및 scFv06475 야생형 및 변이체 둘 모두에 대해 수행하였다. 도 32에 제시된 바와 같이, scFv08168에서, 적중치는 야생형 scFv08168과 대부분 대등한 EC50을 보였고, 이중 돌연변이체 D1을 포함하여 몇몇 돌연변이체는 야생형보다 약간 더 큰 활성을 보였다. 이것은 도 32에 제시된 바와 같이 본원 (물질 및 방법, 섹션 8)에서 설명한 바와 같이 수행된 프로테온 친화도 측정 및 STF 세포 기반 검정 활성으로 확인되었다. scFv08168 변이체의 옥텟 (Octet)에 의한 친화도 순위는 이들이 야생형과 대등함을 보여주었다 (데이터 미제시). 프로테온 동역학 분석에서, D1의 KD는 2.55 nM이고, 야생형의 KD는 3.82 nM이었다 (도 32).
scFv06475에서는, ELISA 및 프로테온 동역학 분석에 의해 검출될 때 활성에 영향을 주는 2개의 돌연변이가 존재하였다. 구체적으로, ELISA에서, VH:G34V 및 VH:I37F의 EC50은 0.76 nM의 야생형에 비해 각각 27 nM 및 4.3 nM인 것으로 밝혀졌다. 프로테온 분석에서, VH:G34V 및 VH:I39F는 오프 레이트의 유의한 감소를 보였다 (데이터 미제시).
이중파라토프
분자의 개선된
열안정성
IgG 기반 융합 분자 902 및 돌연변이체 버전 902T에 대해, 제1 Tm 피크는 47℃로부터 62℃로 이동하였다. 상기 피크는 scFv08168 비폴딩에 대응하였다. 제2 피크는 72℃에서 76℃로 이동하였다. 상기 피크는 도 34에 도시된 바와 같이 Fab06475에 대응하였다.
논의
서열 분석 및 상동성 모델링을 기초로 한 합리적 설계는 scFv에 대한 열안정성 돌연변이를 생성하였다. 본 실시예들에서, 적중률은 scFv08168 및 scFv06475 둘 모두에 대해 약 18%이었다. 가장 유의한 개선은 야생형에 비해 scFv08168에서의 단일 점 돌연변이 VH:I34M에 의한 Tm의 7.5℃ 증가이었다. 서열 관점에서, 상기 위치는 Met으로 고도로 보존되었다. 구조적으로, 상기 위치의 보다 큰 소수성 측쇄는 상기 잔기 주위의 충전을 개선할 수 있다. scFv08168 변이체에서 또 다른 점 돌연변이 VH:S49A는 Tm을 5℃ 상승시켰다. 상기 돌연변이를 상동성 모델링에 의해 선발하였다. 상기 위치는 Ala이 아니라 Ser으로 보다 잘 보존되지만, 구조적으로는 Ala이 상기 잔기 주위의 극성 측쇄의 결여 때문에 보다 양호하게 적합할 수 있다.
상동성 모델링을 이용한 구조 기반 돌연변이 설계를 위해, 메카니즘의 조합을 이용하였다. scFv06475 및 scFv08168에서, 양성 적중치는 다음과 같은 5가지의 생물물리적 고려사항 각각에 의해 발견되었다: 충전 개선, 보다 많은 파이-파이 적층, 보다 많은 수소 결합, 보다 많은 염 가교 및 묻힌 극성기의 제거. 도 29 및 30에 나열된 바와 같이 총 10개의 안정화 돌연변이는 상기 메카니즘의 조합에 의해 확인되었다.
열 안정성을 개선하는 것으로 확인된 돌연변이의 조합 ("유익한 돌연변이")은 이들이 상이한 영역에 위치할 경우 열안정성을 추가로 개선한다. 이것은 scFv08168의 경우에 입증되었다. 유익한 돌연변이 VH:I34M 및 VH:S49A를 조합할 때, Tm은 야생형의 49℃에 비해 62.5℃로 추가로 증가하였다. 이것은 야생형보다 13.5℃가 증가한 것이고, 개별 돌연변이 VH:I34M 및 VH:S49A는 각각 Tm을 7.5℃ 및 5℃ 증가시켰다. 이것은 상가적 효과를 분명히 나타내는 것이었다.
I34M은 scFv08168의 CDR1-H에 매우 가깝지만, 돌연변이는 많은 검정에서 증명된 바와 같이 결합 친화도에 영향을 주지 않았다. CDR은 전체 scFv 또는 전체 항체 안정성에서 일정 기능을 수행한다. 유의한 개선은 돌연변이가 결합 친화도 및 특이성에 영향을 주지 않는 한, 개시된 방법을 이용하여 CDR 영역에 근접한 잔기의 조작을 통해 달성할 수 있다.
대부분의 안정화 돌연변이는 VH 상에 위치하였다. 도 29 및 30에 나열된 15개의 안정화 돌연변이 중에서, 11개는 VH 도메인 상의 돌연변이인 반면, 단지 4개만 VL 도메인 상에 존재하였다.
IgG 또는 다른 포맷 (예를 들어 혈청 알부민 융합체)에 혼입될 경우, 안정화된 VH 및 VL은 분자의 열안정성을 크게 개선하였다. IgG 융합을 위해, 47℃의 보다 낮은 Tm은 scFv08168의 Tm에 대응하고, 72.5℃에서의 보다 높은 피크는 CH2 및 Fab06475의 Tm에 대응한다. 2개의 돌연변이 VH:I34M 및 VH:S49A의 혼입은 scFv08168의 Tm을 47℃에서 62℃로 개선한 반면, 6475 내의 VH:M95F의 혼입은 Fab의 Tm을 72.5℃에서 76℃로 추가로 개선하였다. 개선은 scFv 자체에 대해서뿐만 아니라, VH 및 VL의 개선된 안정성에 의해 Fab에 대해서도 나타났다. 이것은 전체 항체 안정성을 개선하기 위한 보다 일반적인 전략을 제공할 수 있다.
이. 콜라이 시스템에서 HTP 스크리닝과 연결된 합리적인 설계는 매우 빠른 회전 시간 및 높은 적중률을 제공하였다. 이. 콜라이로부터의 물질을 사용하여 측정된 Tm은 포유동물로부터의 것과 관련이 있거나, 또는 순위는 동일하게 유지되었다. 이것은 이. 콜라이에서 HTP로서 실행되는 스크리닝 과정을 크게 단순화시켰다. 플레이트 기반 돌연변이 유발, 형질전환, 발현 및 정제는 1주 미만으로 시간을 단축하였다. 본원에 개시된 방법을 이용하여, 많은 돌연변이가 scFv 또는 다른 항원 결합 단편에서 이루어지고, 열 안정성에 대해 스크리닝될 수 있다. 이어서, 상기 돌연변이된 scFv 또는 항원 결합 단편은 보다 큰 항체 구축물에 상기 안정성을 부여하기 위해 보다 큰 항체 구축물, 예컨대 이중파라토프 항체의 성분으로서 사용될 수 있다.
SEQUENCE LISTING
<110> Ettenberg, Seth Alexander
Cong, Feng
Hartlepp, Felix
Klagge, Ingo Markus
<120> Compositions and Methods of Use for
Therapeutic Low Density Lipoprotein - Related Protein 6
(LRP6) Antibodies
<130> PAT054171-WO-PCT
<150> 61/331985
<151> 2010-05-06
<160> 208
<170> FastSEQ for Windows Version 4.0
<210> 1
<211> 5
<212> PRT
<213> Homo Sapien
<400> 1
Asp Tyr Val Ile Asn
1 5
<210> 2
<211> 17
<212> PRT
<213> Homo Sapien
<400> 2
Gly Ile Ser Trp Ser Gly Val Asn Thr His Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 3
<211> 15
<212> PRT
<213> Homo Sapien
<400> 3
Leu Gly Ala Thr Ala Asn Asn Ile Arg Tyr Lys Phe Met Asp Val
1 5 10 15
<210> 4
<211> 10
<212> PRT
<213> Homo Sapien
<400> 4
Ser Gly Asp Ser Leu Arg Asn Lys Val Tyr
1 5 10
<210> 5
<211> 6
<212> PRT
<213> Homo Sapien
<400> 5
Lys Asn Asn Arg Pro Ser
1 5
<210> 6
<211> 10
<212> PRT
<213> Homo Sapien
<400> 6
Gln Ser Tyr Asp Gly Gln Lys Ser Leu Val
1 5 10
<210> 7
<211> 7
<212> PRT
<213> Homo Sapien
<400> 7
Gly Phe Thr Phe Ser Asp Tyr
1 5
<210> 8
<211> 6
<212> PRT
<213> Homo Sapien
<400> 8
Ser Trp Ser Gly Val Asn
1 5
<210> 9
<211> 15
<212> PRT
<213> Homo Sapien
<400> 9
Leu Gly Ala Thr Ala Asn Asn Ile Arg Tyr Lys Phe Met Asp Val
1 5 10 15
<210> 10
<211> 6
<212> PRT
<213> Homo Sapien
<400> 10
Asp Ser Leu Arg Asn Lys
1 5
<210> 11
<211> 2
<212> PRT
<213> Homo Sapien
<400> 11
Lys Asn
1
<210> 12
<211> 7
<212> PRT
<213> Homo Sapien
<400> 12
Tyr Asp Gly Gln Lys Ser Leu
1 5
<210> 13
<211> 105
<212> PRT
<213> Homo Sapien
<400> 13
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Ser Cys Ser Gly Asp Ser Leu Arg Asn Lys Val Tyr
20 25 30
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr Lys
35 40 45
Asn Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser Asn Ser
50 55 60
Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu Asp Glu
65 70 75 80
Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Gly Gln Lys Ser Leu Val Phe
85 90 95
Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 14
<211> 124
<212> PRT
<213> Homo Sapien
<400> 14
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Val Ile Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Ser Gly Val Asn Thr His Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Gly Ala Thr Ala Asn Asn Ile Arg Tyr Lys Phe Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 15
<211> 315
<212> DNA
<213> Homo Sapien
<400> 15
gatatcgaac tgacccagcc gccttcagtg agcgttgcac caggtcagac cgcgcgtatc 60
tcgtgtagcg gcgattctct tcgtaataag gtttattggt accagcagaa acccgggcag 120
gcgccagttc ttgtgattta taagaataat cgtccctcag gcatcccgga acgctttagc 180
ggatccaaca gcggcaacac cgcgaccctg accattagcg gcactcaggc ggaagacgaa 240
gcggattatt attgccagtc ttatgatggt cagaagtctc ttgtgtttgg cggcggcacg 300
aagttaaccg tccta 315
<210> 16
<211> 372
<212> DNA
<213> Homo Sapien
<400> 16
caggtgcaat tggtggaaag cggcggcggc ctggtgcaac cgggcggcag cctgcgtctg 60
agctgcgcgg cctccggatt taccttttct gattatgtta ttaattgggt gcgccaagcc 120
cctgggaagg gtctcgagtg ggtgagcggt atttcttggt ctggtgttaa tactcattat 180
gctgattctg ttaagggtcg ttttaccatt tcacgtgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgtcttggt 300
gctactgcta ataatattcg ttataagttt atggatgttt ggggccaagg caccctggtg 360
acggttagct ca 372
<210> 17
<211> 211
<212> PRT
<213> Homo Sapien
<400> 17
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Ser Cys Ser Gly Asp Ser Leu Arg Asn Lys Val Tyr
20 25 30
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr Lys
35 40 45
Asn Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser Asn Ser
50 55 60
Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu Asp Glu
65 70 75 80
Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Gly Gln Lys Ser Leu Val Phe
85 90 95
Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln Pro Lys Ala Ala Pro
100 105 110
Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn Lys
115 120 125
Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val Thr
130 135 140
Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu Thr
145 150 155 160
Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser Tyr
165 170 175
Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser Cys
180 185 190
Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro Thr
195 200 205
Glu Cys Ser
210
<210> 18
<211> 454
<212> PRT
<213> Homo Sapien
<400> 18
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Val Ile Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Ser Gly Val Asn Thr His Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Gly Ala Thr Ala Asn Asn Ile Arg Tyr Lys Phe Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
115 120 125
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
130 135 140
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
145 150 155 160
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
165 170 175
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
180 185 190
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
195 200 205
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro
210 215 220
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
225 230 235 240
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
290 295 300
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
305 310 315 320
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
325 330 335
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
340 345 350
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
355 360 365
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
370 375 380
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
385 390 395 400
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
405 410 415
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
420 425 430
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
435 440 445
Ser Leu Ser Pro Gly Lys
450
<210> 19
<211> 633
<212> DNA
<213> Homo Sapien
<400> 19
gatatcgaac tgacccagcc gccttcagtg agcgttgcac caggtcagac cgcgcgtatc 60
tcgtgtagcg gcgattctct tcgtaataag gtttattggt accagcagaa acccgggcag 120
gcgccagttc ttgtgattta taagaataat cgtccctcag gcatcccgga acgctttagc 180
ggatccaaca gcggcaacac cgcgaccctg accattagcg gcactcaggc ggaagacgaa 240
gcggattatt attgccagtc ttatgatggt cagaagtctc ttgtgtttgg cggcggcacg 300
aagttaaccg tcctaggtca gcccaaggct gccccctcgg tcactctgtt cccgccctcc 360
tctgaggagc ttcaagccaa caaggccaca ctggtgtgtc tcataagtga cttctacccg 420
ggagccgtga cagtggcctg gaaggcagat agcagccccg tcaaggcggg agtggagacc 480
accacaccct ccaaacaaag caacaacaag tacgcggcca gcagctatct gagcctgacg 540
cctgagcagt ggaagtccca cagaagctac agctgccagg tcacgcatga agggagcacc 600
gtggagaaga cagtggcccc tacagaatgt tca 633
<210> 20
<211> 1362
<212> DNA
<213> Homo Sapien
<400> 20
caggtgcaat tggtggaaag cggcggcggc ctggtgcaac cgggcggcag cctgcgtctg 60
agctgcgcgg cctccggatt taccttttct gattatgtta ttaattgggt gcgccaagcc 120
cctgggaagg gtctcgagtg ggtgagcggt atttcttggt ctggtgttaa tactcattat 180
gctgattctg ttaagggtcg ttttaccatt tcacgtgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgtcttggt 300
gctactgcta ataatattcg ttataagttt atggatgttt ggggccaagg caccctggtg 360
acggttagct cagcctccac caagggtcca tcggtcttcc ccctggcacc ctcctccaag 420
agcacctctg ggggcacagc ggccctgggc tgcctggtca aggactactt ccccgaaccg 480
gtgacggtgt cgtggaactc aggcgccctg accagcggcg tgcacacctt cccggctgtc 540
ctacagtcct caggactcta ctccctcagc agcgtggtga ccgtgccctc cagcagcttg 600
ggcacccaga cctacatctg caacgtgaat cacaagccca gcaacaccaa ggtggacaag 660
agagttgagc ccaaatcttg tgacaaaact cacacatgcc caccgtgccc agcacctgaa 720
gcagcggggg gaccgtcagt cttcctcttc cccccaaaac ccaaggacac cctcatgatc 780
tcccggaccc ctgaggtcac atgcgtggtg gtggacgtga gccacgaaga ccctgaggtc 840
aagttcaact ggtacgtgga cggcgtggag gtgcataatg ccaagacaaa gccgcgggag 900
gagcagtaca acagcacgta ccgggtggtc agcgtcctca ccgtcctgca ccaggactgg 960
ctgaatggca aggagtacaa gtgcaaggtc tccaacaaag ccctcccagc ccccatcgag 1020
aaaaccatct ccaaagccaa agggcagccc cgagaaccac aggtgtacac cctgccccca 1080
tcccgggagg agatgaccaa gaaccaggtc agcctgacct gcctggtcaa aggcttctat 1140
cccagcgaca tcgccgtgga gtgggagagc aatgggcagc cggagaacaa ctacaagacc 1200
acgcctcccg tgctggactc cgacggctcc ttcttcctct acagcaagct caccgtggac 1260
aagagcaggt ggcagcaggg gaacgtcttc tcatgctccg tgatgcatga ggctctgcac 1320
aaccactaca cgcagaagag cctctccctg tctccgggta aa 1362
<210> 21
<211> 5
<212> PRT
<213> Homo Sapien
<400> 21
Val Asn Gly Met His
1 5
<210> 22
<211> 16
<212> PRT
<213> Homo Sapien
<400> 22
Val Ile Asp Gly Met Gly His Thr Tyr Tyr Ala Asp Ser Val Lys Gly
1 5 10 15
<210> 23
<211> 11
<212> PRT
<213> Homo Sapien
<400> 23
Tyr Asp Tyr Ile Lys Tyr Gly Ala Phe Asp Pro
1 5 10
<210> 24
<211> 11
<212> PRT
<213> Homo Sapien
<400> 24
Ser Gly Asp Asn Ile Gly Ser Lys Tyr Val His
1 5 10
<210> 25
<211> 7
<212> PRT
<213> Homo Sapien
<400> 25
Gly Asp Ser Asn Arg Pro Ser
1 5
<210> 26
<211> 10
<212> PRT
<213> Homo Sapien
<400> 26
Thr Arg Thr Ser Thr Pro Ile Ser Gly Val
1 5 10
<210> 27
<211> 7
<212> PRT
<213> Homo Sapien
<400> 27
Gly Phe Thr Phe Ser Val Asn
1 5
<210> 28
<211> 5
<212> PRT
<213> Homo Sapien
<400> 28
Asp Gly Met Gly His
1 5
<210> 29
<211> 11
<212> PRT
<213> Homo Sapien
<400> 29
Tyr Asp Tyr Ile Lys Tyr Gly Ala Phe Asp Pro
1 5 10
<210> 30
<211> 7
<212> PRT
<213> Homo Sapien
<400> 30
Asp Asn Ile Gly Ser Lys Tyr
1 5
<210> 31
<211> 3
<212> PRT
<213> Homo Sapien
<400> 31
Gly Asp Ser
1
<210> 32
<211> 7
<212> PRT
<213> Homo Sapien
<400> 32
Thr Ser Thr Pro Ile Ser Gly
1 5
<210> 33
<211> 107
<212> PRT
<213> Homo Sapien
<400> 33
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Ser Cys Ser Gly Asp Asn Ile Gly Ser Lys Tyr Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Gly Asp Ser Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Thr Arg Thr Ser Thr Pro Ile Ser Gly
85 90 95
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 34
<211> 119
<212> PRT
<213> Homo Sapien
<400> 34
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Val Asn
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Asp Gly Met Gly His Thr Tyr Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Tyr Asp Tyr Ile Lys Tyr Gly Ala Phe Asp Pro Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 35
<211> 107
<212> PRT
<213> Homo Sapien
<400> 35
Ser Tyr Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Ser Ile Thr Cys Ser Gly Asp Asn Ile Gly Ser Lys Tyr Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ser Pro Val Leu Val Ile Tyr
35 40 45
Gly Asp Ser Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Met
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Thr Arg Thr Ser Thr Pro Ile Ser Gly
85 90 95
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 36
<211> 119
<212> PRT
<213> Homo Sapien
<400> 36
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Val Asn
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Asp Gly Met Gly His Thr Tyr Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Tyr Asp Tyr Ile Lys Tyr Gly Ala Phe Asp Pro Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 37
<211> 321
<212> DNA
<213> Homo Sapien
<400> 37
gatatcgaac tgacccagcc gccttcagtg agcgttgcac caggtcagac cgcgcgtatc 60
tcgtgtagcg gcgataatat tggttctaag tatgttcatt ggtaccagca gaaacccggg 120
caggcgccag ttcttgtgat ttatggtgat tctaatcgtc cctcaggcat cccggaacgc 180
tttagcggat ccaacagcgg caacaccgcg accctgacca ttagcggcac tcaggcggaa 240
gacgaagcgg attattattg cactcgtact tctactccta tttctggtgt gtttggcggc 300
ggcacgaagt taaccgttct t 321
<210> 38
<211> 357
<212> DNA
<213> Homo Sapien
<400> 38
caggtgcaat tggtggaaag cggcggcggc ctggtgcaac cgggcggcag cctgcgtctg 60
agctgcgcgg cctccggatt taccttttct gttaatggta tgcattgggt gcgccaagcc 120
cctgggaagg gtctcgagtg ggtgagcgtt attgatggta tgggtcatac ttattatgct 180
gattctgtta agggtcgttt taccatttca cgtgataatt cgaaaaacac cctgtatctg 240
caaatgaaca gcctgcgtgc ggaagatacg gccgtgtatt attgcgcgcg ttatgattat 300
attaagtatg gtgcttttga tccttggggc caaggcaccc tggtgacggt tagctca 357
<210> 39
<211> 212
<212> PRT
<213> Homo Sapien
<400> 39
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Ser Cys Ser Gly Asp Asn Ile Gly Ser Lys Tyr Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Gly Asp Ser Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Thr Arg Thr Ser Thr Pro Ile Ser Gly
85 90 95
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln Pro Lys Ala
100 105 110
Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala
115 120 125
Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala
130 135 140
Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val
145 150 155 160
Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser
165 170 175
Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr
180 185 190
Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala
195 200 205
Pro Thr Glu Ala
210
<210> 40
<211> 221
<212> PRT
<213> Homo Sapien
<400> 40
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Val Asn
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Asp Gly Met Gly His Thr Tyr Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Tyr Asp Tyr Ile Lys Tyr Gly Ala Phe Asp Pro Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser
210 215 220
<210> 41
<211> 636
<212> DNA
<213> Homo Sapien
<400> 41
gatatcgaac tgacccagcc gccttcagtg agcgttgcac caggtcagac cgcgcgtatc 60
tcgtgtagcg gcgataatat tggttctaag tatgttcatt ggtaccagca gaaacccggg 120
caggcgccag ttcttgtgat ttatggtgat tctaatcgtc cctcaggcat cccggaacgc 180
tttagcggat ccaacagcgg caacaccgcg accctgacca ttagcggcac tcaggcggaa 240
gacgaagcgg attattattg cactcgtact tctactccta tttctggtgt gtttggcggc 300
ggcacgaagt taaccgttct tggccagccg aaagccgcac cgagtgtgac gctgtttccg 360
ccgagcagcg aagaattgca ggcgaacaaa gcgaccctgg tgtgcctgat tagcgacttt 420
tatccgggag ccgtgacagt ggcctggaag gcagatagca gccccgtcaa ggcgggagtg 480
gagaccacca caccctccaa acaaagcaac aacaagtacg cggccagcag ctatctgagc 540
ctgacgcctg agcagtggaa gtcccacaga agctacagct gccaggtcac gcatgagggg 600
agcaccgtgg aaaaaaccgt tgcgccgact gaggcc 636
<210> 42
<211> 663
<212> DNA
<213> Homo Sapien
<400> 42
caggtgcaat tggtggaaag cggcggcggc ctggtgcaac cgggcggcag cctgcgtctg 60
agctgcgcgg cctccggatt taccttttct gttaatggta tgcattgggt gcgccaagcc 120
cctgggaagg gtctcgagtg ggtgagcgtt attgatggta tgggtcatac ttattatgct 180
gattctgtta agggtcgttt taccatttca cgtgataatt cgaaaaacac cctgtatctg 240
caaatgaaca gcctgcgtgc ggaagatacg gccgtgtatt attgcgcgcg ttatgattat 300
attaagtatg gtgcttttga tccttggggc caaggcaccc tggtgacggt tagctcagcg 360
tcgaccaaag gtccaagcgt gtttccgctg gctccgagca gcaaaagcac cagcggcggc 420
acggctgccc tgggctgcct ggttaaagat tatttcccgg aaccagtcac cgtgagctgg 480
aacagcgggg cgctgaccag cggcgtgcat acctttccgg cggtgctgca aagcagcggc 540
ctgtatagcc tgagcagcgt tgtgaccgtg ccgagcagca gcttaggcac tcagacctat 600
atttgcaacg tgaaccataa accgagcaac accaaagtgg ataaaaaagt ggaaccgaaa 660
agc 663
<210> 43
<211> 107
<212> PRT
<213> Homo Sapien
<400> 43
Ser Tyr Glu Leu Thr Gln Pro Leu Ser Val Ser Val Ala Leu Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Gly Gly Asp Asn Ile Gly Ser Lys Tyr Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Gly Asp Ser Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Ala Gln Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Thr Arg Thr Ser Thr Pro Ile Ser Gly
85 90 95
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 44
<211> 119
<212> PRT
<213> Homo Sapien
<400> 44
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Val Asn
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Asp Gly Met Gly His Thr Tyr Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Tyr Asp Tyr Ile Lys Tyr Gly Ala Phe Asp Pro Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 45
<211> 321
<212> DNA
<213> Homo Sapien
<400> 45
agctatgaac tgacccagcc gctgtctgtg agcgtggcgc tgggccagac cgcgcgtatt 60
acctgcggtg gcgataacat tggcagcaaa tatgtgcatt ggtatcagca gaaaccgggc 120
caggcgccgg tgctggtgat ttatggcgat agcaaccgtc cgagcggcat tccggaacgt 180
tttagcggca gcaacagcgg caacaccgcg accctgacca tttctcgcgc gcaggcgggt 240
gatgaagcgg attattattg cacccgtacc agcaccccga ttagcggcgt gtttggcggc 300
ggtacgaagt taaccgttct t 321
<210> 46
<211> 357
<212> DNA
<213> Homo Sapien
<400> 46
gaggtgcaat tgctggaaag cggcggcggc ctggtgcaac cgggcggcag cctgcgtctg 60
agctgcgcgg cctccggatt taccttttct gttaatggta tgcattgggt gcgccaagcc 120
cctgggaagg gtctcgagtg ggtgagcgtt attgatggta tgggtcatac ttattatgct 180
gattctgtta agggtcgttt taccatttca cgtgataatt cgaaaaacac cctgtatctg 240
caaatgaaca gcctgcgtgc ggaagatacg gccgtgtatt attgcgcgcg ttatgattat 300
attaagtatg gtgcttttga tccttggggc caaggcaccc tggtgacggt tagctca 357
<210> 47
<211> 5
<212> PRT
<213> Homo Sapien
<400> 47
Asp Tyr Ala Ile His
1 5
<210> 48
<211> 17
<212> PRT
<213> Homo Sapien
<400> 48
Gly Ile Ser Tyr Ser Gly Ser Ser Thr His Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 49
<211> 14
<212> PRT
<213> Homo Sapien
<400> 49
Gly Ser His Gly Asn Ile Met Ala Lys Arg Tyr Phe Asp Phe
1 5 10
<210> 50
<211> 11
<212> PRT
<213> Homo Sapien
<400> 50
Ser Gly Asp Asn Ile Arg Lys Lys Tyr Val Tyr
1 5 10
<210> 51
<211> 7
<212> PRT
<213> Homo Sapien
<400> 51
Glu Asp Ser Lys Arg Pro Ser
1 5
<210> 52
<211> 11
<212> PRT
<213> Homo Sapien
<400> 52
Ser Thr Ala Asp Ser Gly Ile Asn Asn Gly Val
1 5 10
<210> 53
<211> 7
<212> PRT
<213> Homo Sapien
<400> 53
Gly Phe Thr Phe Ser Asp Tyr
1 5
<210> 54
<211> 6
<212> PRT
<213> Homo Sapien
<400> 54
Ser Tyr Ser Gly Ser Ser
1 5
<210> 55
<211> 14
<212> PRT
<213> Homo Sapien
<400> 55
Gly Ser His Gly Asn Ile Met Ala Lys Arg Tyr Phe Asp Phe
1 5 10
<210> 56
<211> 7
<212> PRT
<213> Homo Sapien
<400> 56
Asp Asn Ile Arg Lys Lys Tyr
1 5
<210> 57
<211> 3
<212> PRT
<213> Homo Sapien
<400> 57
Glu Asp Ser
1
<210> 58
<211> 8
<212> PRT
<213> Homo Sapien
<400> 58
Ala Asp Ser Gly Ile Asn Asn Gly
1 5
<210> 59
<211> 108
<212> PRT
<213> Homo Sapien
<400> 59
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Ser Cys Ser Gly Asp Asn Ile Arg Lys Lys Tyr Val
20 25 30
Tyr Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Glu Asp Ser Lys Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Ser Thr Ala Asp Ser Gly Ile Asn Asn
85 90 95
Gly Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 60
<211> 123
<212> PRT
<213> Homo Sapien
<400> 60
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Ala Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Tyr Ser Gly Ser Ser Thr His Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ser His Gly Asn Ile Met Ala Lys Arg Tyr Phe Asp Phe
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 61
<211> 108
<212> PRT
<213> Homo Sapien
<400> 61
Ser Tyr Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Ser Ile Thr Cys Ser Gly Asp Asn Ile Arg Lys Lys Tyr Val
20 25 30
Tyr Trp Tyr Gln Gln Lys Pro Gly Gln Ser Pro Val Leu Val Ile Tyr
35 40 45
Glu Asp Ser Lys Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Met
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Ser Thr Ala Asp Ser Gly Ile Asn Asn
85 90 95
Gly Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 62
<211> 123
<212> PRT
<213> Homo Sapien
<400> 62
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Ala Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Tyr Ser Gly Ser Ser Thr His Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ser His Gly Asn Ile Met Ala Lys Arg Tyr Phe Asp Phe
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 63
<211> 324
<212> DNA
<213> Homo Sapien
<400> 63
gatatcgaac tgacccagcc gccttcagtg agcgttgcac caggtcagac cgcgcgtatc 60
tcgtgtagcg gcgataatat tcgtaagaag tatgtttatt ggtaccagca gaaacccggg 120
caggcgccag ttcttgtgat ttatgaggat tctaagcgtc cctcaggcat cccggaacgc 180
tttagcggat ccaacagcgg caacaccgcg accctgacca ttagcggcac tcaggcggaa 240
gacgaagcgg attattattg ctctactgct gattctggta ttaataatgg tgtgtttggc 300
ggcggcacga agttaaccgt tctt 324
<210> 64
<211> 369
<212> DNA
<213> Homo Sapien
<400> 64
caggtgcaat tggtggaaag cggcggcggc ctggtgcaac cgggcggcag cctgcgtctg 60
agctgcgcgg cctccggatt taccttttct gattatgcta ttcattgggt gcgccaagcc 120
cctgggaagg gtctcgagtg ggtgagcggt atctcttatt ctggtagctc tacccattat 180
gcggatagcg tgaaaggccg ttttaccatt tcacgtgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgtggttct 300
catggtaata ttatggctaa gcgttatttt gatttttggg gccaaggcac cctggtgacg 360
gttagctca 369
<210> 65
<211> 214
<212> PRT
<213> Homo Sapien
<400> 65
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Ser Cys Ser Gly Asp Asn Ile Arg Lys Lys Tyr Val
20 25 30
Tyr Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Glu Asp Ser Lys Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Ser Thr Ala Asp Ser Gly Ile Asn Asn
85 90 95
Gly Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln Pro Lys
100 105 110
Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln
115 120 125
Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly
130 135 140
Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly
145 150 155 160
Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala
165 170 175
Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser
180 185 190
Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val
195 200 205
Ala Pro Thr Glu Cys Ser
210
<210> 66
<211> 453
<212> PRT
<213> Homo Sapien
<400> 66
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Ala Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Tyr Ser Gly Ser Ser Thr His Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ser His Gly Asn Ile Met Ala Lys Arg Tyr Phe Asp Phe
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly
115 120 125
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
130 135 140
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
145 150 155 160
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
165 170 175
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
180 185 190
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
195 200 205
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys
210 215 220
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala
225 230 235 240
Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
245 250 255
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
260 265 270
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
275 280 285
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
290 295 300
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
305 310 315 320
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
325 330 335
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
340 345 350
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
355 360 365
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
370 375 380
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
385 390 395 400
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
405 410 415
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
420 425 430
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
435 440 445
Leu Ser Pro Gly Lys
450
<210> 67
<211> 642
<212> DNA
<213> Homo Sapien
<400> 67
gatatcgaac tgacccagcc gccttcagtg agcgttgcac caggtcagac cgcgcgtatc 60
tcgtgtagcg gcgataatat tcgtaagaag tatgtttatt ggtaccagca gaaacccggg 120
caggcgccag ttcttgtgat ttatgaggat tctaagcgtc cctcaggcat cccggaacgc 180
tttagcggat ccaacagcgg caacaccgcg accctgacca ttagcggcac tcaggcggaa 240
gacgaagcgg attattattg ctctactgct gattctggta ttaataatgg tgtgtttggc 300
ggcggcacga agttaaccgt cctaggtcag cccaaggctg ccccctcggt cactctgttc 360
ccgccctcct ctgaggagct tcaagccaac aaggccacac tggtgtgtct cataagtgac 420
ttctacccgg gagccgtgac agtggcctgg aaggcagata gcagccccgt caaggcggga 480
gtggagacca ccacaccctc caaacaaagc aacaacaagt acgcggccag cagctatctg 540
agcctgacgc ctgagcagtg gaagtcccac agaagctaca gctgccaggt cacgcatgaa 600
gggagcaccg tggagaagac agtggcccct acagaatgtt ca 642
<210> 68
<211> 1359
<212> DNA
<213> Homo Sapien
<400> 68
caggtgcaat tggtggaaag cggcggcggc ctggtgcaac cgggcggcag cctgcgtctg 60
agctgcgcgg cctccggatt taccttttct gattatgcta ttcattgggt gcgccaagcc 120
cctgggaagg gtctcgagtg ggtgagcggt atctcttatt ctggtagctc tacccattat 180
gcggatagcg tgaaaggccg ttttaccatt tcacgtgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgtggttct 300
catggtaata ttatggccaa gcgttatttt gatttttggg gccaaggcac cctggtgacg 360
gttagctcag cctccaccaa gggtccatcg gtcttccccc tggcaccctc ctccaagagc 420
acctctgggg gcacagcggc cctgggctgc ctggtcaagg actacttccc cgaaccggtg 480
acggtgtcgt ggaactcagg cgccctgacc agcggcgtgc acaccttccc ggctgtccta 540
cagtcctcag gactctactc cctcagcagc gtggtgaccg tgccctccag cagcttgggc 600
acccagacct acatctgcaa cgtgaatcac aagcccagca acaccaaggt ggacaagaga 660
gttgagccca aatcttgtga caaaactcac acatgcccac cgtgcccagc acctgaagca 720
gcggggggac cgtcagtctt cctcttcccc ccaaaaccca aggacaccct catgatctcc 780
cggacccctg aggtcacatg cgtggtggtg gacgtgagcc acgaagaccc tgaggtcaag 840
ttcaactggt acgtggacgg cgtggaggtg cataatgcca agacaaagcc gcgggaggag 900
cagtacaaca gcacgtaccg ggtggtcagc gtcctcaccg tcctgcacca ggactggctg 960
aatggcaagg agtacaagtg caaggtctcc aacaaagccc tcccagcccc catcgagaaa 1020
accatctcca aagccaaagg gcagccccga gaaccacagg tgtacaccct gcccccatcc 1080
cgggaggaga tgaccaagaa ccaggtcagc ctgacctgcc tggtcaaagg cttctatccc 1140
agcgacatcg ccgtggagtg ggagagcaat gggcagccgg agaacaacta caagaccacg 1200
cctcccgtgc tggactccga cggctccttc ttcctctaca gcaagctcac cgtggacaag 1260
agcaggtggc agcaggggaa cgtcttctca tgctccgtga tgcatgaggc tctgcacaac 1320
cactacacgc agaagagcct ctccctgtct ccgggtaaa 1359
<210> 69
<211> 7
<212> PRT
<213> Homo Sapien
<400> 69
Asn Arg Gly Gly Gly Val Gly
1 5
<210> 70
<211> 16
<212> PRT
<213> Homo Sapien
<400> 70
Trp Ile Asp Trp Asp Asp Asp Lys Ser Tyr Ser Thr Ser Leu Lys Thr
1 5 10 15
<210> 71
<211> 9
<212> PRT
<213> Homo Sapien
<400> 71
Met His Leu Pro Leu Val Phe Asp Ser
1 5
<210> 72
<211> 12
<212> PRT
<213> Homo Sapien
<400> 72
Arg Ala Ser Gln Phe Ile Gly Ser Arg Tyr Leu Ala
1 5 10
<210> 73
<211> 7
<212> PRT
<213> Homo Sapien
<400> 73
Gly Ala Ser Asn Arg Ala Thr
1 5
<210> 74
<211> 9
<212> PRT
<213> Homo Sapien
<400> 74
Gln Gln Tyr Tyr Asp Tyr Pro Gln Thr
1 5
<210> 75
<211> 9
<212> PRT
<213> Homo Sapien
<400> 75
Gly Phe Ser Leu Ser Asn Arg Gly Gly
1 5
<210> 76
<211> 5
<212> PRT
<213> Homo Sapien
<400> 76
Asp Trp Asp Asp Asp
1 5
<210> 77
<211> 9
<212> PRT
<213> Homo Sapien
<400> 77
Met His Leu Pro Leu Val Phe Asp Ser
1 5
<210> 78
<211> 8
<212> PRT
<213> Homo Sapien
<400> 78
Ser Gln Phe Ile Gly Ser Arg Tyr
1 5
<210> 79
<211> 3
<212> PRT
<213> Homo Sapien
<400> 79
Gly Ala Ser
1
<210> 80
<211> 6
<212> PRT
<213> Homo Sapien
<400> 80
Tyr Tyr Asp Tyr Pro Gln
1 5
<210> 81
<211> 108
<212> PRT
<213> Homo Sapien
<400> 81
Asp Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Phe Ile Gly Ser Arg
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Asn Arg Ala Thr Gly Val Pro Ala Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Tyr Pro
85 90 95
Gln Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 82
<211> 119
<212> PRT
<213> Homo Sapien
<400> 82
Gln Val Gln Leu Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Asn Arg
20 25 30
Gly Gly Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala Trp Ile Asp Trp Asp Asp Asp Lys Ser Tyr Ser Thr Ser
50 55 60
Leu Lys Thr Arg Leu Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Met His Leu Pro Leu Val Phe Asp Ser Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 83
<211> 324
<212> DNA
<213> Homo Sapien
<400> 83
gatatcgtgc tgacccagag cccggcgacc ctgagcctgt ctccgggcga acgtgcgacc 60
ctgagctgca gagcgagcca gtttattggt tctcgttatc tggcttggta ccagcagaaa 120
ccaggtcaag caccgcgtct attaatttat ggtgcttcta atcgtgcaac tggggtcccg 180
gcgcgtttta gcggctctgg atccggcacg gattttaccc tgaccattag cagcctggaa 240
cctgaagact ttgcgactta ttattgccag cagtattatg attatcctca gacctttggc 300
cagggtacga aagttgaaat taaa 324
<210> 84
<211> 357
<212> DNA
<213> Homo Sapien
<400> 84
caggtgcaat tgaaagaaag cggcccggcc ctggtgaaac cgacccaaac cctgaccctg 60
acctgtacct tttccggatt tagcctgtct aatcgtggtg gtggtgtggg ttggattcgc 120
cagccgcctg ggaaagccct cgagtggctg gcttggatcg attgggatga tgataagtct 180
tatagcacca gcctgaaaac gcgtctgacc attagcaaag atacttcgaa aaatcaggtg 240
gtgctgacta tgaccaacat ggacccggtg gatacggcca cctattattg cgcgcgtatg 300
catcttcctc ttgtttttga ttcttggggc caaggcaccc tggtgacggt tagctca 357
<210> 85
<211> 215
<212> PRT
<213> Homo Sapien
<400> 85
Asp Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Phe Ile Gly Ser Arg
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Asn Arg Ala Thr Gly Val Pro Ala Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Tyr Pro
85 90 95
Gln Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala
100 105 110
Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser
115 120 125
Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu
130 135 140
Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser
145 150 155 160
Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu
165 170 175
Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val
180 185 190
Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys
195 200 205
Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 86
<211> 449
<212> PRT
<213> Homo Sapien
<400> 86
Gln Val Gln Leu Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Asn Arg
20 25 30
Gly Gly Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala Trp Ile Asp Trp Asp Asp Asp Lys Ser Tyr Ser Thr Ser
50 55 60
Leu Lys Thr Arg Leu Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Met His Leu Pro Leu Val Phe Asp Ser Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys
210 215 220
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro
225 230 235 240
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
245 250 255
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
260 265 270
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
275 280 285
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
290 295 300
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
305 310 315 320
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
325 330 335
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
340 345 350
Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr
355 360 365
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
370 375 380
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
385 390 395 400
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
405 410 415
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
420 425 430
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
435 440 445
Lys
<210> 87
<211> 645
<212> DNA
<213> Homo Sapien
<400> 87
gatatcgtgc tgacccagag cccggcgacc ctgagcctgt ctccgggcga acgtgcgacc 60
ctgagctgca gagcgagcca gtttattggt tctcgttatc tggcttggta ccagcagaaa 120
ccaggtcaag caccgcgtct attaatttat ggtgcttcta atcgtgcaac tggggtcccg 180
gcgcgtttta gcggctctgg atccggcacg gattttaccc tgaccattag cagcctggaa 240
cctgaagact ttgcgactta ttattgccag cagtattatg attatcctca gacctttggc 300
cagggtacga aagttgaaat taaacgtacg gtggctgcac catctgtctt catcttcccg 360
ccatctgatg agcagttgaa atctggaact gcctctgttg tgtgcctgct gaataacttc 420
tatcccagag aggccaaagt acagtggaag gtggataacg ccctccaatc gggtaactcc 480
caggagagtg tcacagagca ggacagcaag gacagcacct acagcctcag cagcaccctg 540
acgctgagca aagcagacta cgagaaacac aaagtctacg cctgcgaagt cacccatcag 600
ggcctgagct cgcccgtcac aaagagcttc aacaggggag agtgt 645
<210> 88
<211> 1347
<212> DNA
<213> Homo Sapien
<400> 88
caggtgcaat tgaaagaaag cggcccggcc ctggtgaaac cgacccaaac cctgaccctg 60
acctgtacct tttccggatt tagcctgtct aatcgtggtg gtggtgtggg ttggattcgc 120
cagccgcctg ggaaagccct cgagtggctg gcttggatcg attgggatga tgataagtct 180
tatagcacca gcctgaaaac gcgtctgacc attagcaaag atacttcgaa aaatcaggtg 240
gtgctgacta tgaccaacat ggacccggtg gatacggcca cctattattg cgcgcgtatg 300
catcttcctc ttgtttttga ttcttggggc caaggcaccc tggtgacggt tagctcagcc 360
tccaccaagg gtccatcggt cttccccctg gcaccctcct ccaagagcac ctctgggggc 420
acagcggccc tgggctgcct ggtcaaggac tacttccccg aaccggtgac ggtgtcgtgg 480
aactcaggcg ccctgaccag cggcgtgcac accttcccgg ctgtcctaca gtcctcagga 540
ctctactccc tcagcagcgt ggtgaccgtg ccctccagca gcttgggcac ccagacctac 600
atctgcaacg tgaatcacaa gcccagcaac accaaggtgg acaagagagt tgagcccaaa 660
tcttgtgaca aaactcacac atgcccaccg tgcccagcac ctgaagcagc ggggggaccg 720
tcagtcttcc tcttcccccc aaaacccaag gacaccctca tgatctcccg gacccctgag 780
gtcacatgcg tggtggtgga cgtgagccac gaagaccctg aggtcaagtt caactggtac 840
gtggacggcg tggaggtgca taatgccaag acaaagccgc gggaggagca gtacaacagc 900
acgtaccggg tggtcagcgt cctcaccgtc ctgcaccagg actggctgaa tggcaaggag 960
tacaagtgca aggtctccaa caaagccctc ccagccccca tcgagaaaac catctccaaa 1020
gccaaagggc agccccgaga accacaggtg tacaccctgc ccccatcccg ggaggagatg 1080
accaagaacc aggtcagcct gacctgcctg gtcaaaggct tctatcccag cgacatcgcc 1140
gtggagtggg agagcaatgg gcagccggag aacaactaca agaccacgcc tcccgtgctg 1200
gactccgacg gctccttctt cctctacagc aagctcaccg tggacaagag caggtggcag 1260
caggggaacg tcttctcatg ctccgtgatg catgaggctc tgcacaacca ctacacgcag 1320
aagagcctct ccctgtctcc gggtaaa 1347
<210> 89
<211> 119
<212> PRT
<213> Homo Sapien
<400> 89
Gln Val Thr Leu Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Asn Arg
20 25 30
Gly Gly Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala Trp Ile Asp Trp Asp Asp Asp Lys Ser Tyr Ser Thr Ser
50 55 60
Leu Lys Thr Arg Leu Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Met His Leu Pro Leu Val Phe Asp Ser Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 90
<211> 108
<212> PRT
<213> Homo Sapien
<400> 90
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Phe Ile Gly Ser Arg
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Tyr Asp Tyr Pro
85 90 95
Gln Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 91
<211> 357
<212> DNA
<213> Homo Sapien
<400> 91
caggtcacac tgaaagagtc cggccctgcc ctggtcaaac ccacccagac cctgaccctg 60
acatgcacct tcagcggctt cagcctgagc aacagaggcg gcggagtggg ctggatcaga 120
cagcctcccg gcaaggccct ggaatggctg gcctggatcg actgggacga cgacaagagc 180
tacagcacca gcctgaaaac ccggctgacc atcagcaagg acaccagcaa gaaccaggtg 240
gtgctgacca tgaccaacat ggaccccgtg gacaccgcca cctactactg cgcccggatg 300
catctgcccc tggtgttcga tagctggggc cagggcaccc tggtcaccgt cagctca 357
<210> 92
<211> 324
<212> DNA
<213> Homo Sapien
<400> 92
gaaatcgtgc tgacccagag ccccgccacc ctgtctctga gccctggcga gagagccacc 60
ctgagctgcc gggccagcca gttcatcggc agcagatacc tggcttggta tcagcagaag 120
cccggccagg cccccagact gctgatctac ggcgccagca accgggccac cggcatccct 180
gccagatttt ctggcagcgg cagcggcacc gacttcaccc tgaccatcag cagcctggaa 240
cccgaggact tcgccgtgta ctactgccag cagtactacg actaccccca gaccttcggc 300
cagggcacca aggtggaaat caag 324
<210> 93
<211> 7
<212> PRT
<213> Homo Sapien
<400> 93
Asn Arg Gly Gly Gly Val Gly
1 5
<210> 94
<211> 16
<212> PRT
<213> Homo Sapien
<400> 94
Trp Ile Asp Trp Asp Asp Asp Lys Ser Tyr Ser Thr Ser Leu Lys Thr
1 5 10 15
<210> 95
<211> 9
<212> PRT
<213> Homo Sapien
<400> 95
Met His Leu Pro Leu Val Phe Asp Ser
1 5
<210> 96
<211> 12
<212> PRT
<213> Homo Sapien
<400> 96
Arg Ala Ser Gln Phe Ile Gly Ser Arg Tyr Leu Ala
1 5 10
<210> 97
<211> 7
<212> PRT
<213> Homo Sapien
<400> 97
Gly Ala Ser Asn Arg Ala Thr
1 5
<210> 98
<211> 9
<212> PRT
<213> Homo Sapien
<400> 98
Gln Gln Tyr Trp Ser Ile Pro Ile Thr
1 5
<210> 99
<211> 9
<212> PRT
<213> Homo Sapien
<400> 99
Gly Phe Ser Leu Ser Asn Arg Gly Gly
1 5
<210> 100
<211> 5
<212> PRT
<213> Homo Sapien
<400> 100
Asp Trp Asp Asp Asp
1 5
<210> 101
<211> 9
<212> PRT
<213> Homo Sapien
<400> 101
Met His Leu Pro Leu Val Phe Asp Ser
1 5
<210> 102
<211> 8
<212> PRT
<213> Homo Sapien
<400> 102
Ser Gln Phe Ile Gly Ser Arg Tyr
1 5
<210> 103
<211> 3
<212> PRT
<213> Homo Sapien
<400> 103
Gly Ala Ser
1
<210> 104
<211> 6
<212> PRT
<213> Homo Sapien
<400> 104
Tyr Trp Ser Ile Pro Ile
1 5
<210> 105
<211> 108
<212> PRT
<213> Homo Sapien
<400> 105
Asp Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Phe Ile Gly Ser Arg
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Asn Arg Ala Thr Gly Val Pro Ala Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Trp Ser Ile Pro
85 90 95
Ile Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 106
<211> 119
<212> PRT
<213> Homo Sapien
<400> 106
Gln Val Gln Leu Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Asn Arg
20 25 30
Gly Gly Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala Trp Ile Asp Trp Asp Asp Asp Lys Ser Tyr Ser Thr Ser
50 55 60
Leu Lys Thr Arg Leu Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Met His Leu Pro Leu Val Phe Asp Ser Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 107
<211> 108
<212> PRT
<213> Homo Sapien
<400> 107
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Phe Ile Gly Ser Arg
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Trp Ser Ile Pro
85 90 95
Ile Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 108
<211> 119
<212> PRT
<213> Homo Sapien
<400> 108
Gln Val Thr Leu Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Asn Arg
20 25 30
Gly Gly Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala Trp Ile Asp Trp Asp Asp Asp Lys Ser Tyr Ser Thr Ser
50 55 60
Leu Lys Thr Arg Leu Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Met His Leu Pro Leu Val Phe Asp Ser Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 109
<211> 324
<212> DNA
<213> Homo Sapien
<400> 109
gatatcgtgc tgacccagag cccggcgacc ctgagcctgt ctccgggcga acgtgcgacc 60
ctgagctgca gagcgagcca gtttattggt tctcgttatc tggcttggta ccagcagaaa 120
ccaggtcaag caccgcgtct attaatttat ggtgcttcta atcgtgcaac tggggtcccg 180
gcgcgtttta gcggctctgg atccggcacg gattttaccc tgaccattag cagcctggaa 240
cctgaagact ttgcggtgta ttattgccag cagtattggt ctattcctat tacctttggc 300
cagggtacga aagttgaaat taaa 324
<210> 110
<211> 357
<212> DNA
<213> Homo Sapien
<400> 110
caggtgcaat tgaaagaaag cggcccggcc ctggtgaaac cgacccaaac cctgaccctg 60
acctgtacct tttccggatt tagcctgtct aatcgtggtg gtggtgtggg ttggattcgc 120
cagccgcctg ggaaagccct cgagtggctg gcttggatcg attgggatga tgataagtct 180
tatagcacca gcctgaaaac gcgtctgacc attagcaaag atacttcgaa aaatcaggtg 240
gtgctgacta tgaccaacat ggacccggtg gatacggcca cctattattg cgcgcgtatg 300
catcttcctc ttgtttttga ttcttggggc caaggcaccc tggtgacggt tagctca 357
<210> 111
<211> 215
<212> PRT
<213> Homo Sapien
<400> 111
Asp Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Phe Ile Gly Ser Arg
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Asn Arg Ala Thr Gly Val Pro Ala Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Trp Ser Ile Pro
85 90 95
Ile Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala
100 105 110
Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser
115 120 125
Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu
130 135 140
Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser
145 150 155 160
Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu
165 170 175
Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val
180 185 190
Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys
195 200 205
Ser Phe Asn Arg Gly Glu Ala
210 215
<210> 112
<211> 221
<212> PRT
<213> Homo Sapien
<400> 112
Gln Val Gln Leu Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Asn Arg
20 25 30
Gly Gly Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala Trp Ile Asp Trp Asp Asp Asp Lys Ser Tyr Ser Thr Ser
50 55 60
Leu Lys Thr Arg Leu Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Met His Leu Pro Leu Val Phe Asp Ser Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser
210 215 220
<210> 113
<211> 645
<212> DNA
<213> Homo Sapien
<400> 113
gatatcgtgc tgacccagag cccggcgacc ctgagcctgt ctccgggcga acgtgcgacc 60
ctgagctgca gagcgagcca gtttattggt tctcgttatc tggcttggta ccagcagaaa 120
ccaggtcaag caccgcgtct attaatttat ggtgcttcta atcgtgcaac tggggtcccg 180
gcgcgtttta gcggctctgg atccggcacg gattttaccc tgaccattag cagcctggaa 240
cctgaagact ttgcggtgta ttattgccag cagtattggt ctattcctat tacctttggc 300
cagggtacga aagttgaaat taaacgtacg gtggctgctc cgagcgtgtt tatttttccg 360
ccgagcgatg aacaactgaa aagcggcacg gcgagcgtgg tgtgcctgct gaacaacttt 420
tatccgcgtg aagcgaaagt tcagtggaaa gtagacaacg cgctgcaaag cggcaacagc 480
caggaaagcg tgaccgaaca ggatagcaaa gatagcacct attctctgag cagcaccctg 540
accctgagca aagcggatta tgaaaaacat aaagtgtatg cgtgcgaagt gacccatcaa 600
ggtctgagca gcccggtgac taaatctttt aatcgtggcg aggcc 645
<210> 114
<211> 663
<212> DNA
<213> Homo Sapien
<400> 114
caggtgcaat tgaaagaaag cggcccggcc ctggtgaaac cgacccaaac cctgaccctg 60
acctgtacct tttccggatt tagcctgtct aatcgtggtg gtggtgtggg ttggattcgc 120
cagccgcctg ggaaagccct cgagtggctg gcttggatcg attgggatga tgataagtct 180
tatagcacca gcctgaaaac gcgtctgacc attagcaaag atacttcgaa aaatcaggtg 240
gtgctgacta tgaccaacat ggacccggtg gatacggcca cctattattg cgcgcgtatg 300
catcttcctc ttgtttttga ttcttggggc caaggcaccc tggtgacggt tagctcagcg 360
tcgaccaaag gtccaagcgt gtttccgctg gctccgagca gcaaaagcac cagcggcggc 420
acggctgccc tgggctgcct ggttaaagat tatttcccgg aaccagtcac cgtgagctgg 480
aacagcgggg cgctgaccag cggcgtgcat acctttccgg cggtgctgca aagcagcggc 540
ctgtatagcc tgagcagcgt tgtgaccgtg ccgagcagca gcttaggcac tcagacctat 600
atttgcaacg tgaaccataa accgagcaac accaaagtgg ataaaaaagt ggaaccgaaa 660
agc 663
<210> 115
<211> 5
<212> PRT
<213> Homo Sapien
<400> 115
Ser Tyr Gly Met Ser
1 5
<210> 116
<211> 17
<212> PRT
<213> Homo Sapien
<400> 116
Asn Ile Ser Asn Asp Gly His Tyr Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 117
<211> 11
<212> PRT
<213> Homo Sapien
<400> 117
Phe Gln Ala Ser Tyr Leu Asp Ile Met Asp Tyr
1 5 10
<210> 118
<211> 11
<212> PRT
<213> Homo Sapien
<400> 118
Ser Gly Asp Asn Ile Gly Ser Lys Tyr Val His
1 5 10
<210> 119
<211> 7
<212> PRT
<213> Homo Sapien
<400> 119
Asn Asp Ser Asn Arg Pro Ser
1 5
<210> 120
<211> 10
<212> PRT
<213> Homo Sapien
<400> 120
Gln Ala Trp Gly Asp Asn Gly Thr Arg Val
1 5 10
<210> 121
<211> 7
<212> PRT
<213> Homo Sapien
<400> 121
Gly Phe Thr Phe Ser Ser Tyr
1 5
<210> 122
<211> 6
<212> PRT
<213> Homo Sapien
<400> 122
Ser Asn Asp Gly His Tyr
1 5
<210> 123
<211> 11
<212> PRT
<213> Homo Sapien
<400> 123
Phe Gln Ala Ser Tyr Leu Asp Ile Met Asp Tyr
1 5 10
<210> 124
<211> 7
<212> PRT
<213> Homo Sapien
<400> 124
Asp Asn Ile Gly Ser Lys Tyr
1 5
<210> 125
<211> 3
<212> PRT
<213> Homo Sapien
<400> 125
Asn Asp Ser
1
<210> 126
<211> 7
<212> PRT
<213> Homo Sapien
<400> 126
Trp Gly Asp Asn Gly Thr Arg
1 5
<210> 127
<211> 107
<212> PRT
<213> Homo Sapien
<400> 127
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Ser Gly Asp Asn Ile Gly Ser Lys Tyr Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Asn Asp Ser Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ala Trp Gly Asp Asn Gly Thr Arg
85 90 95
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 128
<211> 120
<212> PRT
<213> Homo Sapien
<400> 128
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Asn Ile Ser Asn Asp Gly His Tyr Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Phe Gln Ala Ser Tyr Leu Asp Ile Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 129
<211> 107
<212> PRT
<213> Homo Sapien
<400> 129
Ser Tyr Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Ser Ile Thr Cys Ser Gly Asp Asn Ile Gly Ser Lys Tyr Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ser Pro Val Leu Val Ile Tyr
35 40 45
Asn Asp Ser Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Met
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ala Trp Gly Asp Asn Gly Thr Arg
85 90 95
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 130
<211> 120
<212> PRT
<213> Homo Sapien
<400> 130
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Asn Ile Ser Asn Asp Gly His Tyr Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Phe Gln Ala Ser Tyr Leu Asp Ile Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 131
<211> 321
<212> DNA
<213> Homo Sapien
<400> 131
gatatcgaac tgacccagcc gccttcagtg agcgttgcac caggtcagag cattaccatc 60
tcgtgtagcg gcgataatat tggttctaag tatgttcatt ggtaccagca gaaacccggg 120
caggcgccag ttcttgtgat ttataatgat tctaatcgtc cctcaggcat cccggaacgc 180
tttagcggat ccaacagcgg caacaccgcg accctgacca ttagcggcac tcaggcggaa 240
gacgaagcgg attattattg ccaggcttgg ggtgataatg gtactcgtgt gtttggcggc 300
ggcacgaagt taaccgttct t 321
<210> 132
<211> 360
<212> DNA
<213> Homo Sapien
<400> 132
caggtgcaat tggtggaaag cggcggcggc ctggtgcaac cgggcggcag cctgcgtctg 60
agctgcgcgg cctccggatt taccttttct tcttatggta tgtcttgggt gcgccaagcc 120
cctgggaagg gtctcgagtg ggtgagcaat atttctaatg atggtcatta tacttattat 180
gctgattctg ttaagggtcg ttttaccatt tcacgtgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgttttcag 300
gcttcttatc ttgatattat ggattattgg ggccaaggca ccctggtgac ggttagctca 360
<210> 133
<211> 212
<212> PRT
<213> Homo Sapien
<400> 133
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Ser Gly Asp Asn Ile Gly Ser Lys Tyr Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Asn Asp Ser Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ala Trp Gly Asp Asn Gly Thr Arg
85 90 95
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln Pro Lys Ala
100 105 110
Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala
115 120 125
Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala
130 135 140
Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val
145 150 155 160
Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser
165 170 175
Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr
180 185 190
Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala
195 200 205
Pro Thr Glu Ala
210
<210> 134
<211> 222
<212> PRT
<213> Homo Sapien
<400> 134
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Asn Ile Ser Asn Asp Gly His Tyr Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Phe Gln Ala Ser Tyr Leu Asp Ile Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser
210 215 220
<210> 135
<211> 636
<212> DNA
<213> Homo Sapien
<400> 135
gatatcgaac tgacccagcc gccttcagtg agcgttgcac caggtcagag cattaccatc 60
tcgtgtagcg gcgataatat tggttctaag tatgttcatt ggtaccagca gaaacccggg 120
caggcgccag ttcttgtgat ttataatgat tctaatcgtc cctcaggcat cccggaacgc 180
tttagcggat ccaacagcgg caacaccgcg accctgacca ttagcggcac tcaggcggaa 240
gacgaagcgg attattattg ccaggcttgg ggtgataatg gtactcgtgt gtttggcggc 300
ggcacgaagt taaccgttct tggccagccg aaagccgcac cgagtgtgac gctgtttccg 360
ccgagcagcg aagaattgca ggcgaacaaa gcgaccctgg tgtgcctgat tagcgacttt 420
tatccgggag ccgtgacagt ggcctggaag gcagatagca gccccgtcaa ggcgggagtg 480
gagaccacca caccctccaa acaaagcaac aacaagtacg cggccagcag ctatctgagc 540
ctgacgcctg agcagtggaa gtcccacaga agctacagct gccaggtcac gcatgagggg 600
agcaccgtgg aaaaaaccgt tgcgccgact gaggcc 636
<210> 136
<211> 666
<212> DNA
<213> Homo Sapien
<400> 136
caggtgcaat tggtggaaag cggcggcggc ctggtgcaac cgggcggcag cctgcgtctg 60
agctgcgcgg cctccggatt taccttttct tcttatggta tgtcttgggt gcgccaagcc 120
cctgggaagg gtctcgagtg ggtgagcaat atttctaatg atggtcatta tacttattat 180
gctgattctg ttaagggtcg ttttaccatt tcacgtgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgttttcag 300
gcttcttatc ttgatattat ggattattgg ggccaaggca ccctggtgac ggttagctca 360
gcgtcgacca aaggtccaag cgtgtttccg ctggctccga gcagcaaaag caccagcggc 420
ggcacggctg ccctgggctg cctggttaaa gattatttcc cggaaccagt caccgtgagc 480
tggaacagcg gggcgctgac cagcggcgtg catacctttc cggcggtgct gcaaagcagc 540
ggcctgtata gcctgagcag cgttgtgacc gtgccgagca gcagcttagg cactcagacc 600
tatatttgca acgtgaacca taaaccgagc aacaccaaag tggataaaaa agtggaaccg 660
aaaagc 666
<210> 137
<211> 107
<212> PRT
<213> Homo Sapien
<400> 137
Ser Tyr Glu Leu Thr Gln Pro Leu Ser Val Ser Val Ala Leu Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Gly Gly Asp Asn Ile Gly Ser Lys Tyr Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Asn Asp Ser Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Ala Gln Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ala Trp Gly Asp Asn Gly Thr Arg
85 90 95
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 138
<211> 120
<212> PRT
<213> Homo Sapien
<400> 138
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Asn Ile Ser Asn Asp Gly His Tyr Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Phe Gln Ala Ser Tyr Leu Asp Ile Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 139
<211> 321
<212> DNA
<213> Homo Sapien
<400> 139
agctatgaac tgacccagcc gctgagtgtt agcgttgcgc tgggtcagac cgcgcgtatt 60
acctgcggcg gtgataacat tggcagcaaa tatgtgcatt ggtatcagca gaaaccgggc 120
caggcgccgg tgctggtgat ttataacgat agcaaccgtc cgagcggcat tccggaacgt 180
tttagcggca gcaacagcgg caataccgcg accctgacca ttagccgtgc gcaggcgggt 240
gatgaagcgg attattattg ccaggcgtgg ggcgataatg gtacgcgtgt gtttggcggt 300
ggtacgaagt taaccgttct t 321
<210> 140
<211> 360
<212> DNA
<213> Homo Sapien
<400> 140
gaggtgcaat tgctggaaag cggcggcggc ctggtgcaac cgggcggcag cctgcgtctg 60
agctgcgcgg cctccggatt taccttttct tcttatggta tgtcttgggt gcgccaagcc 120
cctgggaagg gtctcgagtg ggtgagcaat atttctaatg atggtcatta tacttattat 180
gctgattctg ttaagggtcg ttttaccatt tcacgtgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgttttcag 300
gcttcttatc ttgatattat ggattattgg ggccaaggca ccctggtgac ggttagctca 360
<210> 141
<211> 726
<212> DNA
<213> Homo Sapien
<400> 141
gatatcgttc tgacccagag tccggcaacc ctgagcctga gtccgggtga acgtgccacc 60
ctgagctgtc gtgcaagcca gtttattggt agccgttatc tggcatggta tcagcagaaa 120
ccgggtcagg caccgcgtct gctgatttat ggtgcaagca atcgtgcaac cggtgttccg 180
gcacgtttta gcggtagcgg tagtggcacc gattttaccc tgaccattag cagcctggaa 240
ccggaagatt ttgcaaccta ttattgccag cagtattatg attatccgca gacctttggt 300
cagggcacca aggtggaaat taaaggtggt ggtggtagcg gtggtggtgg ctcaggtggt 360
ggcggtagtc aggttcaatt gaaagaaagc ggtccggcac tggttaaacc gacccagacc 420
ctgaccctga catgtacctt tagcggtttt agcctgagca atcgtggtgg tggtgttggt 480
tggattcgtc agcctccggg taaagcactg gaatggctgg catggattga ttgggatgat 540
gataaaagct atagcaccag cctgaaaacc cgtctgacca ttagtaaaga taccagcaaa 600
aatcaggtgg ttctgaccat gaccaatatg gatccggttg ataccgccac ctattattgt 660
gcacgtatgc atctgccgct ggtttttgat agctggggtc agggtacact agttaccgtt 720
agcagc 726
<210> 142
<211> 242
<212> PRT
<213> Homo Sapien
<400> 142
Asp Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Phe Ile Gly Ser Arg
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Asn Arg Ala Thr Gly Val Pro Ala Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Tyr Pro
85 90 95
Gln Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly
100 105 110
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Lys
115 120 125
Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln Thr Leu Thr Leu Thr
130 135 140
Cys Thr Phe Ser Gly Phe Ser Leu Ser Asn Arg Gly Gly Gly Val Gly
145 150 155 160
Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu Trp Leu Ala Trp Ile
165 170 175
Asp Trp Asp Asp Asp Lys Ser Tyr Ser Thr Ser Leu Lys Thr Arg Leu
180 185 190
Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Val Val Leu Thr Met Thr
195 200 205
Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr Cys Ala Arg Met His
210 215 220
Leu Pro Leu Val Phe Asp Ser Trp Gly Gln Gly Thr Leu Val Thr Val
225 230 235 240
Ser Ser
<210> 143
<211> 741
<212> DNA
<213> Homo Sapien
<400> 143
gatatcgtgc tgacacagag ccctgccacc ctgtctctga gccctggcga gagagccacc 60
ctgagctgcc gggccagcca gttcatcggc tcccgctacc tggcctggta tcagcagaag 120
cccggacagg ctcccagact gctgatctac ggcgccagca acagagctac cggcgtgccc 180
gccagatttt ctggcagcgg cagcggcacc gacttcaccc tgaccatcag cagcctggaa 240
cccgaggact tcgccaccta ctactgccag cagtactacg actaccccca gaccttcggc 300
cagggcacca aggtggagat caagggcgga ggcggatccg ggggtggcgg aagtggaggc 360
ggaggaagcg gagggggcgg aagccaggtg caattgaaag agtccggccc tgccctggtg 420
aagcctaccc agaccctgac cctgacatgc accttcagcg gcttcagcct gagcaacaga 480
ggcggcggag tgggctggat cagacagcct cccggcaagg ccctggaatg gctggcctgg 540
atcgactggg acgacgacaa gagctacagc accagcctga aaacccggct gaccatctcc 600
aaggacacca gcaagaacca ggtggtgctc accatgacca acatggaccc cgtggacacc 660
gccacctatt attgcgcccg gatgcatctg cccctggtgt tcgatagctg gggccaggga 720
accctggtga cagtgtccag c 741
<210> 144
<211> 247
<212> PRT
<213> Homo Sapien
<400> 144
Asp Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Phe Ile Gly Ser Arg
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Asn Arg Ala Thr Gly Val Pro Ala Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Tyr Pro
85 90 95
Gln Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly
100 105 110
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gln Val Gln Leu Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln
130 135 140
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Asn Arg
145 150 155 160
Gly Gly Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
165 170 175
Trp Leu Ala Trp Ile Asp Trp Asp Asp Asp Lys Ser Tyr Ser Thr Ser
180 185 190
Leu Lys Thr Arg Leu Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Val
195 200 205
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
210 215 220
Cys Ala Arg Met His Leu Pro Leu Val Phe Asp Ser Trp Gly Gln Gly
225 230 235 240
Thr Leu Val Thr Val Ser Ser
245
<210> 145
<211> 726
<212> DNA
<213> Homo Sapien
<400> 145
caggttcaat tgaaagaaag cggtccggca ctggttaaac cgacccagac cctgaccctg 60
acatgtacct ttagcggttt tagcctgagc aatcgtggtg gtggtgttgg ttggattcgt 120
cagcctccgg gtaaagcact ggaatggctg gcatggattg attgggatga tgataaaagc 180
tatagcacca gcctgaaaac ccgtctgacc attagcaaag ataccagcaa aaatcaggtt 240
gttctgacca tgaccaatat ggatccggtt gataccgcaa cctattattg tgcacgtatg 300
catctgccgc tggtttttga tagctggggt cagggtacac tagttaccgt tagcagcggt 360
ggtggtggta gcggtggtgg cggttcaggt ggtggtggca gtgatatcgt tctgacccag 420
agtccggcaa ccctgagcct gagtccgggt gaacgtgcca ccctgagctg tcgtgcaagc 480
cagtttattg gtagccgtta tctggcatgg tatcagcaga aaccgggtca ggcaccgcgt 540
ctgctgattt atggtgcaag caatcgtgca accggtgttc cggcacgttt tagcggtagc 600
ggtagtggca ccgattttac cctgaccatt agtagcctgg aaccggaaga ttttgccacc 660
tattattgcc agcagtatta tgattatccg cagacctttg gtcagggcac caaggtggaa 720
attaaa 726
<210> 146
<211> 242
<212> PRT
<213> Homo Sapien
<400> 146
Gln Val Gln Leu Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Asn Arg
20 25 30
Gly Gly Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala Trp Ile Asp Trp Asp Asp Asp Lys Ser Tyr Ser Thr Ser
50 55 60
Leu Lys Thr Arg Leu Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Met His Leu Pro Leu Val Phe Asp Ser Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Asp Ile Val Leu Thr Gln Ser Pro Ala Thr
130 135 140
Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser
145 150 155 160
Gln Phe Ile Gly Ser Arg Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly
165 170 175
Gln Ala Pro Arg Leu Leu Ile Tyr Gly Ala Ser Asn Arg Ala Thr Gly
180 185 190
Val Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
195 200 205
Thr Ile Ser Ser Leu Glu Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln
210 215 220
Gln Tyr Tyr Asp Tyr Pro Gln Thr Phe Gly Gln Gly Thr Lys Val Glu
225 230 235 240
Ile Lys
<210> 147
<211> 741
<212> DNA
<213> Homo Sapien
<400> 147
caggttcaat tgaaagaaag cggtccggca ctggttaaac cgacccagac cctgaccctg 60
acatgtacct ttagcggttt tagcctgagc aatcgtggtg gtggtgttgg ttggattcgt 120
cagcctccgg gtaaagcact ggaatggctg gcatggattg attgggatga tgataaaagc 180
tatagcacca gcctgaaaac ccgtctgacc attagcaaag ataccagcaa aaatcaggtt 240
gttctgacca tgaccaatat ggatccggtt gataccgcaa cctattattg tgcacgtatg 300
catctgccgc tggtttttga tagctggggt cagggtacac tagttaccgt tagcagcggt 360
ggtggtggta gcggtggtgg cggttcaggt ggtggtggca gtggcggtgg tggtagtgat 420
atcgttctga cccagagtcc ggcaaccctg agcctgagtc cgggtgaacg tgccaccctg 480
agctgtcgtg caagccagtt tattggtagc cgttatctgg catggtatca gcagaaaccg 540
ggtcaggcac cgcgtctgct gatttatggt gcaagcaatc gtgcaaccgg tgttccggca 600
cgttttagcg gtagcggtag tggcaccgat tttaccctga ccattagtag cctggaaccg 660
gaagattttg ccacctatta ttgccagcag tattatgatt atccgcagac ctttggtcag 720
ggcaccaagg tggaaattaa a 741
<210> 148
<211> 247
<212> PRT
<213> Homo Sapien
<400> 148
Gln Val Gln Leu Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Asn Arg
20 25 30
Gly Gly Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala Trp Ile Asp Trp Asp Asp Asp Lys Ser Tyr Ser Thr Ser
50 55 60
Leu Lys Thr Arg Leu Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Met His Leu Pro Leu Val Phe Asp Ser Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Val Leu Thr
130 135 140
Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu
145 150 155 160
Ser Cys Arg Ala Ser Gln Phe Ile Gly Ser Arg Tyr Leu Ala Trp Tyr
165 170 175
Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Gly Ala Ser
180 185 190
Asn Arg Ala Thr Gly Val Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly
195 200 205
Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu Asp Phe Ala
210 215 220
Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Tyr Pro Gln Thr Phe Gly Gln
225 230 235 240
Gly Thr Lys Val Glu Ile Lys
245
<210> 149
<211> 732
<212> DNA
<213> Homo Sapien
<400> 149
gatatcgaac tgacccagcc tccgagcgtt agcgttgcac cgggtcagac cgcacgtatt 60
agctgtagcg gtgatagcct gcgtaataaa gtttattggt atcagcagaa accgggtcag 120
gcaccggttc tggttattta taaaaataat cgtccgagcg gtattccgga acgttttagc 180
ggtagcaata gcggtaatac cgcaaccctg accattagcg gcacccaggc agaagatgaa 240
gcagattatt attgccagag ctatgatggt cagaaaagcc tggtttttgg tggtggcacc 300
aagcttaccg ttctgggtgg tggtggtagc ggtggtggtg gctcaggtgg tggcggttct 360
caggttcaat tggttgaaag tggtggtggt ctggttcagc ctggtggtag cctgcgtctg 420
agctgtgcag caagcggttt tacctttagc gattatgtga ttaattgggt tcgccaggca 480
ccgggtaaag gtctggaatg ggttagcggt attagctggt caggtgttaa tacccattat 540
gcagatagcg tgaaaggtcg ttttaccatt agccgtgata atagcaaaaa taccctgtat 600
ctgcagatga atagcctgcg tgcagaagat accgcagttt attattgtgc acgtctgggt 660
gcaaccgcaa ataatattcg ctataaattt atggatgtgt ggggtcaggg tacactagtt 720
accgttagca gc 732
<210> 150
<211> 244
<212> PRT
<213> Homo Sapien
<400> 150
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Ser Cys Ser Gly Asp Ser Leu Arg Asn Lys Val Tyr
20 25 30
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr Lys
35 40 45
Asn Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser Asn Ser
50 55 60
Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu Asp Glu
65 70 75 80
Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Gly Gln Lys Ser Leu Val Phe
85 90 95
Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly Ser Gly Gly
100 105 110
Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Val Glu Ser Gly
115 120 125
Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala
130 135 140
Ser Gly Phe Thr Phe Ser Asp Tyr Val Ile Asn Trp Val Arg Gln Ala
145 150 155 160
Pro Gly Lys Gly Leu Glu Trp Val Ser Gly Ile Ser Trp Ser Gly Val
165 170 175
Asn Thr His Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg
180 185 190
Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala
195 200 205
Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg Leu Gly Ala Thr Ala Asn
210 215 220
Asn Ile Arg Tyr Lys Phe Met Asp Val Trp Gly Gln Gly Thr Leu Val
225 230 235 240
Thr Val Ser Ser
<210> 151
<211> 747
<212> DNA
<213> Homo Sapien
<400> 151
gatatcgaac tgacccagcc tccgagcgtt agcgttgcac cgggtcagac cgcacgtatt 60
agctgtagcg gtgatagcct gcgtaataaa gtttattggt atcagcagaa accgggtcag 120
gcaccggttc tggttattta taaaaataat cgtccgagcg gtattccgga acgttttagc 180
ggtagcaata gcggtaatac cgcaaccctg accattagcg gcacccaggc agaagatgaa 240
gcagattatt attgccagag ctatgatggt cagaaaagcc tggtttttgg tggtggcacc 300
aagcttaccg ttctgggtgg tggtggtagc ggtggtggtg gctcaggtgg tggcggttct 360
ggtggcggtg gttcacaggt tcaattggtt gaaagtggtg gtggtctggt tcagcctggt 420
ggtagcctgc gtctgagctg tgcagcaagc ggttttacct ttagcgatta tgtgattaat 480
tgggttcgcc aggcaccggg taaaggtctg gaatgggtta gcggtattag ctggtcaggt 540
gttaataccc attatgcaga tagcgtgaaa ggtcgtttta ccattagccg tgataatagc 600
aaaaataccc tgtatctgca gatgaatagc ctgcgtgcag aagataccgc agtttattat 660
tgtgcacgtc tgggtgcaac cgcaaataat attcgctata aatttatgga tgtgtggggt 720
cagggtacac tagttaccgt tagcagc 747
<210> 152
<211> 249
<212> PRT
<213> Homo Sapien
<400> 152
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Ser Cys Ser Gly Asp Ser Leu Arg Asn Lys Val Tyr
20 25 30
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr Lys
35 40 45
Asn Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser Asn Ser
50 55 60
Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu Asp Glu
65 70 75 80
Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Gly Gln Lys Ser Leu Val Phe
85 90 95
Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly Ser Gly Gly
100 105 110
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln
115 120 125
Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg
130 135 140
Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr Val Ile Asn
145 150 155 160
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser Gly Ile
165 170 175
Ser Trp Ser Gly Val Asn Thr His Tyr Ala Asp Ser Val Lys Gly Arg
180 185 190
Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met
195 200 205
Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg Leu
210 215 220
Gly Ala Thr Ala Asn Asn Ile Arg Tyr Lys Phe Met Asp Val Trp Gly
225 230 235 240
Gln Gly Thr Leu Val Thr Val Ser Ser
245
<210> 153
<211> 732
<212> DNA
<213> Homo Sapien
<400> 153
caggttcaat tggttgaaag cggtggtggt ctggttcagc ctggtggtag cctgcgtctg 60
agctgtgcag caagcggttt tacctttagc gattatgtga ttaattgggt tcgtcaggca 120
ccgggtaaag gtctggaatg ggttagcggt attagctggt caggtgttaa tacccattat 180
gcagatagcg tgaaaggtcg ttttaccatt agccgtgata atagcaaaaa taccctgtat 240
ctgcagatga atagcctgcg tgcagaagat accgcagttt attattgtgc acgtctgggt 300
gcaaccgcaa ataatattcg ctataaattt atggatgtgt ggggtcaggg tacactagtt 360
accgttagca gtggtggtgg tggtagcggt ggtggcggat ctggtggcgg tggcagtgat 420
atcgaactga cccagcctcc gagcgttagc gttgcaccgg gtcagaccgc acgtattagc 480
tgtagcggtg atagtctgcg taataaagtt tattggtatc agcagaaacc gggtcaggct 540
ccggttctgg ttatttataa aaataatcgt ccgagcggta ttccggaacg ttttagcggt 600
agcaatagcg gtaataccgc aaccctgacc attagcggca cccaggcaga agatgaagcc 660
gattattatt gtcagagcta tgatggtcag aaaagcctgg tttttggtgg tggcaccaag 720
cttaccgttc tg 732
<210> 154
<211> 244
<212> PRT
<213> Homo Sapien
<400> 154
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Val Ile Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Ser Gly Val Asn Thr His Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Gly Ala Thr Ala Asn Asn Ile Arg Tyr Lys Phe Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Glu Leu Thr
130 135 140
Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln Thr Ala Arg Ile Ser
145 150 155 160
Cys Ser Gly Asp Ser Leu Arg Asn Lys Val Tyr Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Gln Ala Pro Val Leu Val Ile Tyr Lys Asn Asn Arg Pro Ser
180 185 190
Gly Ile Pro Glu Arg Phe Ser Gly Ser Asn Ser Gly Asn Thr Ala Thr
195 200 205
Leu Thr Ile Ser Gly Thr Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys
210 215 220
Gln Ser Tyr Asp Gly Gln Lys Ser Leu Val Phe Gly Gly Gly Thr Lys
225 230 235 240
Leu Thr Val Leu
<210> 155
<211> 747
<212> DNA
<213> Homo Sapien
<400> 155
caggttcaat tggttgaaag cggtggtggt ctggttcagc ctggtggtag cctgcgtctg 60
agctgtgcag caagcggttt tacctttagc gattatgtga ttaattgggt tcgtcaggca 120
ccgggtaaag gtctggaatg ggttagcggt attagctggt caggtgttaa tacccattat 180
gcagatagcg tgaaaggtcg ttttaccatt agccgtgata atagcaaaaa taccctgtat 240
ctgcagatga atagcctgcg tgcagaagat accgcagttt attattgtgc acgtctgggt 300
gcaaccgcaa ataatattcg ctataaattt atggatgtgt ggggtcaggg tacactagtt 360
accgttagca gtggtggtgg tggtagcggt ggtggcggat ctggtggcgg tggttcaggt 420
ggtggtggca gtgatatcga actgacccag cctccgagcg ttagcgttgc accgggtcag 480
accgcacgta ttagctgtag cggtgatagt ctgcgtaata aagtttattg gtatcagcag 540
aaaccgggtc aggctccggt tctggttatt tataaaaata atcgtccgag cggtattccg 600
gaacgtttta gcggtagcaa tagcggtaat accgcaaccc tgaccattag cggcacccag 660
gcagaagatg aagccgatta ttattgtcag agctatgatg gtcagaaaag cctggttttt 720
ggtggtggca ccaagcttac cgttctg 747
<210> 156
<211> 249
<212> PRT
<213> Homo Sapien
<400> 156
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Val Ile Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Ser Gly Val Asn Thr His Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Gly Ala Thr Ala Asn Asn Ile Arg Tyr Lys Phe Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
130 135 140
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
145 150 155 160
Thr Ala Arg Ile Ser Cys Ser Gly Asp Ser Leu Arg Asn Lys Val Tyr
165 170 175
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr Lys
180 185 190
Asn Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser Asn Ser
195 200 205
Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu Asp Glu
210 215 220
Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Gly Gln Lys Ser Leu Val Phe
225 230 235 240
Gly Gly Gly Thr Lys Leu Thr Val Leu
245
<210> 157
<211> 723
<212> DNA
<213> Homo Sapien
<400> 157
gatatcgaac tgacccagcc tccgagcgtt agcgttgcac cgggtcagac cgcacgtatt 60
agctgtagcg gtgataatat tggcagcaaa tatgtgcatt ggtatcagca gaaaccgggt 120
caggcaccgg ttctggttat ttatggtgat agcaatcgtc cgagcggtat tccggaacgt 180
tttagcggta gcaatagcgg taataccgca accctgacca ttagcggcac ccaggcagaa 240
gatgaagcag attattattg tacccgtacc agcaccccga ttagcggtgt ttttggtggt 300
ggcaccaagc ttaccgttct gggtggtggt ggtagcggtg gtggtggctc aggtggtggt 360
ggttcacagg ttcaattggt tgaaagtggt ggtggtctgg ttcagcctgg tggtagcctg 420
cgtctgagct gtgcagcaag cggttttacc tttagcgtta atggtatgca ttgggttcgc 480
caggcaccgg gtaaaggtct ggaatgggtt agcgttattg atggtatggg ccatacctat 540
tatgccgata gcgttaaagg tcgttttacc attagccgtg ataatagcaa aaataccctg 600
tatctgcaga tgaatagcct gcgtgcagaa gataccgcag tttattattg cgcacgctat 660
gattatatta aatatggtgc ctttgatccg tggggtcagg gtacactagt taccgttagc 720
agc 723
<210> 158
<211> 241
<212> PRT
<213> Homo Sapien
<400> 158
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Ser Cys Ser Gly Asp Asn Ile Gly Ser Lys Tyr Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Gly Asp Ser Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Thr Arg Thr Ser Thr Pro Ile Ser Gly
85 90 95
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Val Glu
115 120 125
Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys
130 135 140
Ala Ala Ser Gly Phe Thr Phe Ser Val Asn Gly Met His Trp Val Arg
145 150 155 160
Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser Val Ile Asp Gly Met
165 170 175
Gly His Thr Tyr Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser
180 185 190
Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg
195 200 205
Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg Tyr Asp Tyr Ile Lys
210 215 220
Tyr Gly Ala Phe Asp Pro Trp Gly Gln Gly Thr Leu Val Thr Val Ser
225 230 235 240
Ser
<210> 159
<211> 738
<212> DNA
<213> Homo Sapien
<400> 159
gatatcgaac tgacccagcc tccgagcgtt agcgttgcac cgggtcagac cgcacgtatt 60
agctgtagcg gtgataatat tggcagcaaa tatgtgcatt ggtatcagca gaaaccgggt 120
caggcaccgg ttctggttat ttatggtgat agcaatcgtc cgagcggtat tccggaacgt 180
tttagcggta gcaatagcgg taataccgca accctgacca ttagcggcac ccaggcagaa 240
gatgaagcag attattattg tacccgtacc agcaccccga ttagcggtgt ttttggtggt 300
ggcaccaagc ttaccgttct gggtggtggt ggtagcggtg gtggtggctc aggtggtggc 360
ggttctggtg gcggtggttc acaggttcaa ttggttgaaa gtggtggtgg tctggttcag 420
cctggtggta gcctgcgtct gagctgtgca gcaagcggtt ttacctttag cgttaatggt 480
atgcattggg ttcgccaggc accgggtaaa ggtctggaat gggttagcgt tattgatggt 540
atgggccata cctattatgc cgatagcgtt aaaggtcgtt ttaccattag ccgtgataat 600
agcaaaaata ccctgtatct gcagatgaat agcctgcgtg cagaagatac cgcagtttat 660
tattgcgcac gctatgatta tattaaatat ggtgcctttg atccgtgggg tcagggtaca 720
ctagttaccg ttagcagc 738
<210> 160
<211> 246
<212> PRT
<213> Homo Sapien
<400> 160
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Ser Cys Ser Gly Asp Asn Ile Gly Ser Lys Tyr Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Gly Asp Ser Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Thr Arg Thr Ser Thr Pro Ile Ser Gly
85 90 95
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln
115 120 125
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
130 135 140
Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Val Asn Gly
145 150 155 160
Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser
165 170 175
Val Ile Asp Gly Met Gly His Thr Tyr Tyr Ala Asp Ser Val Lys Gly
180 185 190
Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln
195 200 205
Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg
210 215 220
Tyr Asp Tyr Ile Lys Tyr Gly Ala Phe Asp Pro Trp Gly Gln Gly Thr
225 230 235 240
Leu Val Thr Val Ser Ser
245
<210> 161
<211> 723
<212> DNA
<213> Homo Sapien
<400> 161
caggttcaat tggttgaaag cggtggtggt ctggttcagc ctggtggtag cctgcgtctg 60
agctgtgcag caagcggttt tacctttagc gttaatggta tgcattgggt tcgtcaggca 120
ccgggtaaag gtctggaatg ggttagcgtt attgatggta tgggccatac ctattatgcc 180
gatagcgtta aaggtcgttt taccattagc cgtgataata gcaaaaatac cctgtatctg 240
cagatgaata gcctgcgtgc agaagatacc gcagtttatt attgtgcccg ttatgattat 300
attaaatatg gtgcctttga tccgtggggt cagggtacac tagttaccgt tagcagtggt 360
ggtggtggta gcggtggtgg cggatctggt ggcggtggtt cagatatcga actgacccag 420
cctccgagcg ttagcgttgc accgggtcag accgcacgta ttagctgtag cggtgataat 480
attggcagca aatatgtgca ttggtatcag cagaaaccgg gtcaggctcc ggttctggtt 540
atttatggtg atagcaatcg tccgagcggt attccggaac gttttagcgg tagcaatagc 600
ggtaataccg caaccctgac cattagcggc acccaggcag aagatgaagc cgattattat 660
tgcacccgta ccagcacccc gattagcggt gtttttggtg gtggcaccaa gcttaccgtt 720
ctg 723
<210> 162
<211> 241
<212> PRT
<213> Homo Sapien
<400> 162
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Val Asn
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Asp Gly Met Gly His Thr Tyr Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Tyr Asp Tyr Ile Lys Tyr Gly Ala Phe Asp Pro Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Asp Ile Glu Leu Thr Gln Pro Pro Ser Val
130 135 140
Ser Val Ala Pro Gly Gln Thr Ala Arg Ile Ser Cys Ser Gly Asp Asn
145 150 155 160
Ile Gly Ser Lys Tyr Val His Trp Tyr Gln Gln Lys Pro Gly Gln Ala
165 170 175
Pro Val Leu Val Ile Tyr Gly Asp Ser Asn Arg Pro Ser Gly Ile Pro
180 185 190
Glu Arg Phe Ser Gly Ser Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile
195 200 205
Ser Gly Thr Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Thr Arg Thr
210 215 220
Ser Thr Pro Ile Ser Gly Val Phe Gly Gly Gly Thr Lys Leu Thr Val
225 230 235 240
Leu
<210> 163
<211> 738
<212> DNA
<213> Homo Sapien
<400> 163
caggttcaat tggttgaaag cggtggtggt ctggttcagc ctggtggtag cctgcgtctg 60
agctgtgcag caagcggttt tacctttagc gttaatggta tgcattgggt tcgtcaggca 120
ccgggtaaag gtctggaatg ggttagcgtt attgatggta tgggccatac ctattatgcc 180
gatagcgtta aaggtcgttt taccattagc cgtgataata gcaaaaatac cctgtatctg 240
cagatgaata gcctgcgtgc agaagatacc gcagtttatt attgtgcccg ttatgattat 300
attaaatatg gtgcctttga tccgtggggt cagggtacac tagttaccgt tagcagtggt 360
ggtggtggta gcggtggtgg cggatctggt ggcggtggtt caggtggtgg tggcagtgat 420
atcgaactga cccagcctcc gagcgttagc gttgcaccgg gtcagaccgc acgtattagc 480
tgtagcggtg ataatattgg cagcaaatat gtgcattggt atcagcagaa accgggtcag 540
gctccggttc tggttattta tggtgatagc aatcgtccga gcggtattcc ggaacgtttt 600
agcggtagca atagcggtaa taccgcaacc ctgaccatta gcggcaccca ggcagaagat 660
gaagccgatt attattgcac ccgtaccagc accccgatta gcggtgtttt tggtggtggc 720
accaagctta ccgttctg 738
<210> 164
<211> 246
<212> PRT
<213> Homo Sapien
<400> 164
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Val Asn
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Asp Gly Met Gly His Thr Tyr Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Tyr Asp Tyr Ile Lys Tyr Gly Ala Phe Asp Pro Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Glu Leu Thr
130 135 140
Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln Thr Ala Arg Ile Ser
145 150 155 160
Cys Ser Gly Asp Asn Ile Gly Ser Lys Tyr Val His Trp Tyr Gln Gln
165 170 175
Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr Gly Asp Ser Asn Arg
180 185 190
Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser Asn Ser Gly Asn Thr
195 200 205
Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu Asp Glu Ala Asp Tyr
210 215 220
Tyr Cys Thr Arg Thr Ser Thr Pro Ile Ser Gly Val Phe Gly Gly Gly
225 230 235 240
Thr Lys Leu Thr Val Leu
245
<210> 165
<211> 2121
<212> DNA
<213> Homo Sapien
<400> 165
caggtgcaat tggtcgagtc tggcggagga ctggtgcagc ctggtggcag cctgagactg 60
agctgcgccg ccagcggctt caccttcagc gactacgtga tcaactgggt gcgacaggcc 120
cctggaaagg gcctggaatg ggtgtccggc atctcttggt ctggcgtgaa cacccactac 180
gccgacagcg tgaagggccg gttcaccatc agccgggaca acagcaagaa caccctgtac 240
ctgcagatga acagcctgag agccgaggac accgccgtgt actactgtgc cagactgggc 300
gccaccgcca acaacatccg gtacaagttc atggacgtgt ggggccaggg cacactggtg 360
accgtcagct cagctagcac caagggcccc agcgtgttcc ccctggcccc cagcagcaag 420
agcaccagcg gcggcacagc cgccctgggc tgcctggtga aggactactt ccccgagccc 480
gtgaccgtgt cctggaacag cggagccctg acctccggcg tgcacacctt ccccgccgtg 540
ctgcagagca gcggcctgta cagcctgtcc agcgtggtga cagtgcccag cagcagcctg 600
ggcacccaga cctacatctg caacgtgaac cacaagccca gcaacaccaa ggtggacaag 660
agagtggagc ccaagagctg cgacaagacc cacacctgcc ccccctgccc agccccagag 720
gcagcgggcg gaccctccgt gttcctgttc ccccccaagc ccaaggacac cctgatgatc 780
agcaggaccc ccgaggtgac ctgcgtggtg gtggacgtga gccacgagga cccagaggtg 840
aagttcaact ggtacgtgga cggcgtggag gtgcacaacg ccaagaccaa gcccagagag 900
gagcagtaca acagcaccta cagggtggtg tccgtgctga ccgtgctgca ccaggactgg 960
ctgaacggca aggaatacaa gtgcaaggtc tccaacaagg ccctgccagc ccccatcgaa 1020
aagaccatca gcaaggccaa gggccagcca cgggagcccc aggtgtacac cctgcccccc 1080
tcccgggagg agatgaccaa gaaccaggtg tccctgacct gtctggtgaa gggcttctac 1140
cccagcgaca tcgccgtgga gtgggagagc aacggccagc ccgagaacaa ctacaagacc 1200
acccccccag tgctggacag cgacggcagc ttcttcctgt acagcaagct gaccgtggac 1260
aagtccaggt ggcagcaggg caacgtgttc agctgcagcg tgatgcacga agcgctgcac 1320
aaccactaca cccagaagag cctgagcctg tcccccggca agggcggctc cggcggaagc 1380
gatatcgtgc tgacacagag ccctgccacc ctgtctctga gccctggcga gagagccacc 1440
ctgagctgcc gggccagcca gttcatcggc tcccgctacc tggcctggta tcagcagaag 1500
cccggacagg ctcccagact gctgatctac ggcgccagca acagagctac cggcgtgccc 1560
gccagatttt ctggcagcgg cagcggcacc gacttcaccc tgaccatcag cagcctggaa 1620
cccgaggact tcgccaccta ctactgccag cagtactacg actaccccca gaccttcggc 1680
cagggcacca aggtggagat caagggcgga ggcggatccg ggggtggcgg aagtggaggc 1740
ggaggaagcg gagggggcgg aagccaggtg caattgaaag agtccggccc tgccctggtg 1800
aagcctaccc agaccctgac cctgacatgc accttcagcg gcttcagcct gagcaacaga 1860
ggcggcggag tgggctggat cagacagcct cccggcaagg ccctggaatg gctggcctgg 1920
atcgactggg acgacgacaa gagctacagc accagcctga aaacccggct gaccatctcc 1980
aaggacacca gcaagaacca ggtggtgctc accatgacca acatggaccc cgtggacacc 2040
gccacctatt attgcgcccg gatgcatctg cccctggtgt tcgatagctg gggccaggga 2100
accctggtga cagtgtccag c 2121
<210> 166
<211> 707
<212> PRT
<213> Homo Sapien
<400> 166
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Val Ile Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Ser Gly Val Asn Thr His Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Gly Ala Thr Ala Asn Asn Ile Arg Tyr Lys Phe Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
115 120 125
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
130 135 140
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
145 150 155 160
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
165 170 175
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
180 185 190
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
195 200 205
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro
210 215 220
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
225 230 235 240
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
290 295 300
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
305 310 315 320
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
325 330 335
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
340 345 350
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
355 360 365
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
370 375 380
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
385 390 395 400
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
405 410 415
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
420 425 430
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
435 440 445
Ser Leu Ser Pro Gly Lys Gly Gly Ser Gly Gly Ser Asp Ile Val Leu
450 455 460
Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr
465 470 475 480
Leu Ser Cys Arg Ala Ser Gln Phe Ile Gly Ser Arg Tyr Leu Ala Trp
485 490 495
Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Gly Ala
500 505 510
Ser Asn Arg Ala Thr Gly Val Pro Ala Arg Phe Ser Gly Ser Gly Ser
515 520 525
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu Asp Phe
530 535 540
Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Tyr Pro Gln Thr Phe Gly
545 550 555 560
Gln Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly
565 570 575
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu
580 585 590
Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln Thr Leu Thr Leu
595 600 605
Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Asn Arg Gly Gly Gly Val
610 615 620
Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu Trp Leu Ala Trp
625 630 635 640
Ile Asp Trp Asp Asp Asp Lys Ser Tyr Ser Thr Ser Leu Lys Thr Arg
645 650 655
Leu Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Val Val Leu Thr Met
660 665 670
Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr Cys Ala Arg Met
675 680 685
His Leu Pro Leu Val Phe Asp Ser Trp Gly Gln Gly Thr Leu Val Thr
690 695 700
Val Ser Ser
705
<210> 167
<211> 105
<212> PRT
<213> Homo Sapien
<400> 167
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Ser Cys Ser Gly Asp Ser Leu Arg Asn Lys Val Tyr
20 25 30
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr Lys
35 40 45
Asn Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser Asn Ser
50 55 60
Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu Asp Glu
65 70 75 80
Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Gly Gln Lys Ser Leu Val Phe
85 90 95
Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 168
<211> 315
<212> DNA
<213> Homo Sapien
<400> 168
gacatcgagc tgacccagcc cccttctgtg tctgtggccc ctggccagac cgccagaatc 60
agctgcagcg gcgacagcct gcggaacaag gtgtactggt atcagcagaa gcccggccag 120
gctcccgtgc tggtgatcta caagaacaac cggcccagcg gcatccctga gcggttcagc 180
ggcagcaaca gcggcaatac cgccaccctg accatcagcg gcacccaggc cgaagatgag 240
gccgactact actgccagag ctacgacggc cagaaaagcc tggtgttcgg cggaggcacc 300
aagcttaccg tgctg 315
<210> 169
<211> 633
<212> DNA
<213> Homo Sapien
<400> 169
gacatcgagc tgacccagcc cccttctgtg tctgtggccc ctggccagac cgccagaatc 60
agctgcagcg gcgacagcct gcggaacaag gtgtactggt atcagcagaa gcccggccag 120
gctcccgtgc tggtgatcta caagaacaac cggcccagcg gcatccctga gcggttcagc 180
ggcagcaaca gcggcaatac cgccaccctg accatcagcg gcacccaggc cgaagatgag 240
gccgactact actgccagag ctacgacggc cagaaaagcc tggtgttcgg cggaggcacc 300
aagcttaccg tgctgggcca gcccaaagcc gcccctagcg tgaccctgtt cccccccagc 360
agcgaggaac tgcaggccaa caaggccacc ctggtctgcc tgatcagcga cttctaccct 420
ggcgccgtga ccgtggcctg gaaggccgac agcagccccg tgaaggccgg cgtggagaca 480
accaccccca gcaagcagag caacaacaag tacgccgcca gcagctacct gagcctgacc 540
cccgagcagt ggaagagcca cagaagctac agctgccagg tcacccacga gggcagcacc 600
gtggagaaaa ccgtggcccc caccgagtgc agc 633
<210> 170
<211> 211
<212> PRT
<213> Homo Sapien
<400> 170
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Ser Cys Ser Gly Asp Ser Leu Arg Asn Lys Val Tyr
20 25 30
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr Lys
35 40 45
Asn Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser Asn Ser
50 55 60
Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu Asp Glu
65 70 75 80
Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Gly Gln Lys Ser Leu Val Phe
85 90 95
Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln Pro Lys Ala Ala Pro
100 105 110
Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn Lys
115 120 125
Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val Thr
130 135 140
Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu Thr
145 150 155 160
Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser Tyr
165 170 175
Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser Cys
180 185 190
Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro Thr
195 200 205
Glu Cys Ser
210
<210> 171
<211> 706
<212> PRT
<213> Homo Sapien
<400> 171
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Val Ile Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Ser Gly Val Asn Thr His Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Gly Ala Thr Ala Asn Asn Ile Arg Tyr Lys Phe Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
115 120 125
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
130 135 140
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
145 150 155 160
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
165 170 175
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
180 185 190
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
195 200 205
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro
210 215 220
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
225 230 235 240
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
290 295 300
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
305 310 315 320
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
325 330 335
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
340 345 350
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
355 360 365
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
370 375 380
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
385 390 395 400
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
405 410 415
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
420 425 430
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
435 440 445
Ser Leu Ser Pro Gly Gly Gly Ser Gly Gly Ser Asp Ile Val Leu Thr
450 455 460
Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu
465 470 475 480
Ser Cys Arg Ala Ser Gln Phe Ile Gly Ser Arg Tyr Leu Ala Trp Tyr
485 490 495
Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Gly Ala Ser
500 505 510
Asn Arg Ala Thr Gly Val Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly
515 520 525
Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu Asp Phe Ala
530 535 540
Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Tyr Pro Gln Thr Phe Gly Gln
545 550 555 560
Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly
565 570 575
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Lys
580 585 590
Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln Thr Leu Thr Leu Thr
595 600 605
Cys Thr Phe Ser Gly Phe Ser Leu Ser Asn Arg Gly Gly Gly Val Gly
610 615 620
Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu Trp Leu Ala Trp Ile
625 630 635 640
Asp Trp Asp Asp Asp Lys Ser Tyr Ser Thr Ser Leu Lys Thr Arg Leu
645 650 655
Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Val Val Leu Thr Met Thr
660 665 670
Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr Cys Ala Arg Met His
675 680 685
Leu Pro Leu Val Phe Asp Ser Trp Gly Gln Gly Thr Leu Val Thr Val
690 695 700
Ser Ser
705
<210> 172
<211> 2118
<212> DNA
<213> Homo Sapien
<400> 172
caggtgcaat tggtcgagtc tggcggagga ctggtgcagc ctggtggcag cctgagactg 60
agctgcgccg ccagcggctt caccttcagc gactacgtga tcaactgggt gcgacaggcc 120
cctggaaagg gcctggaatg ggtgtccggc atctcttggt ctggcgtgaa cacccactac 180
gccgacagcg tgaagggccg gttcaccatc agccgggaca acagcaagaa caccctgtac 240
ctgcagatga acagcctgag agccgaggac accgccgtgt actactgtgc cagactgggc 300
gccaccgcca acaacatccg gtacaagttc atggacgtgt ggggccaggg cacactggtg 360
accgtcagct cagctagcac caagggcccc agcgtgttcc ccctggcccc cagcagcaag 420
agcaccagcg gcggcacagc cgccctgggc tgcctggtga aggactactt ccccgagccc 480
gtgaccgtgt cctggaacag cggagccctg acctccggcg tgcacacctt ccccgccgtg 540
ctgcagagca gcggcctgta cagcctgtcc agcgtggtga cagtgcccag cagcagcctg 600
ggcacccaga cctacatctg caacgtgaac cacaagccca gcaacaccaa ggtggacaag 660
agagtggagc ccaagagctg cgacaagacc cacacctgcc ccccctgccc agccccagag 720
gcagcgggcg gaccctccgt gttcctgttc ccccccaagc ccaaggacac cctgatgatc 780
agcaggaccc ccgaggtgac ctgcgtggtg gtggacgtga gccacgagga cccagaggtg 840
aagttcaact ggtacgtgga cggcgtggag gtgcacaacg ccaagaccaa gcccagagag 900
gagcagtaca acagcaccta cagggtggtg tccgtgctga ccgtgctgca ccaggactgg 960
ctgaacggca aggaatacaa gtgcaaggtc tccaacaagg ccctgccagc ccccatcgaa 1020
aagaccatca gcaaggccaa gggccagcca cgggagcccc aggtgtacac cctgcccccc 1080
tcccgggagg agatgaccaa gaaccaggtg tccctgacct gtctggtgaa gggcttctac 1140
cccagcgaca tcgccgtgga gtgggagagc aacggccagc ccgagaacaa ctacaagacc 1200
acccccccag tgctggacag cgacggcagc ttcttcctgt acagcaagct gaccgtggac 1260
aagtccaggt ggcagcaggg caacgtgttc agctgcagcg tgatgcacga agcgctgcac 1320
aaccactaca cccagaagag cctgagcctg tcccccggcg gcggctccgg cggaagcgat 1380
atcgtgctga cacagagccc tgccaccctg tctctgagcc ctggcgagag agccaccctg 1440
agctgccggg ccagccagtt catcggctcc cgctacctgg cctggtatca gcagaagccc 1500
ggacaggctc ccagactgct gatctacggc gccagcaaca gagctaccgg cgtgcccgcc 1560
agattttctg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctggaaccc 1620
gaggacttcg ccacctacta ctgccagcag tactacgact acccccagac cttcggccag 1680
ggcaccaagg tggagatcaa gggcggaggc ggatccgggg gtggcggaag tggaggcgga 1740
ggaagcggag ggggcggaag ccaggtgcaa ttgaaagagt ccggccctgc cctggtgaag 1800
cctacccaga ccctgaccct gacatgcacc ttcagcggct tcagcctgag caacagaggc 1860
ggcggagtgg gctggatcag acagcctccc ggcaaggccc tggaatggct ggcctggatc 1920
gactgggacg acgacaagag ctacagcacc agcctgaaaa cccggctgac catctccaag 1980
gacaccagca agaaccaggt ggtgctcacc atgaccaaca tggaccccgt ggacaccgcc 2040
acctattatt gcgcccggat gcatctgccc ctggtgttcg atagctgggg ccagggaacc 2100
ctggtgacag tgtccagc 2118
<210> 173
<211> 707
<212> PRT
<213> Homo Sapien
<400> 173
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Val Ile Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Ser Gly Val Asn Thr His Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Gly Ala Thr Ala Asn Asn Ile Arg Tyr Lys Phe Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
115 120 125
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
130 135 140
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
145 150 155 160
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
165 170 175
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
180 185 190
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
195 200 205
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro
210 215 220
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
225 230 235 240
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
290 295 300
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
305 310 315 320
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
325 330 335
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
340 345 350
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
355 360 365
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
370 375 380
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
385 390 395 400
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
405 410 415
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
420 425 430
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
435 440 445
Ser Leu Ser Pro Gly Lys Gly Gly Ser Gly Gly Ser Asp Ile Val Leu
450 455 460
Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr
465 470 475 480
Leu Ser Cys Arg Ala Ser Gln Phe Ile Gly Ser Arg Tyr Leu Ala Trp
485 490 495
Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Gly Ala
500 505 510
Ser Asn Arg Ala Thr Gly Val Pro Ala Arg Phe Ser Gly Ser Gly Ser
515 520 525
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu Asp Phe
530 535 540
Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Tyr Pro Gln Thr Phe Gly
545 550 555 560
Gln Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly
565 570 575
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu
580 585 590
Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln Thr Leu Thr Leu
595 600 605
Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Asn Arg Gly Gly Gly Val
610 615 620
Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu Trp Leu Ala Trp
625 630 635 640
Ile Asp Trp Asp Asp Asp Lys Ser Tyr Ser Thr Ser Leu Lys Thr Arg
645 650 655
Leu Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Val Val Leu Thr Met
660 665 670
Thr Asn Met Asp Ala Val Asp Thr Ala Thr Tyr Tyr Cys Ala Arg Met
675 680 685
His Leu Pro Leu Val Phe Asp Ser Trp Gly Gln Gly Thr Leu Val Thr
690 695 700
Val Ser Ser
705
<210> 174
<211> 2121
<212> DNA
<213> Homo Sapien
<400> 174
caggtgcaat tggtcgagtc tggcggagga ctggtgcagc ctggtggcag cctgagactg 60
agctgcgccg ccagcggctt caccttcagc gactacgtga tcaactgggt gcgacaggcc 120
cctggaaagg gcctggaatg ggtgtccggc atctcttggt ctggcgtgaa cacccactac 180
gccgacagcg tgaagggccg gttcaccatc agccgggaca acagcaagaa caccctgtac 240
ctgcagatga acagcctgag agccgaggac accgccgtgt actactgtgc cagactgggc 300
gccaccgcca acaacatccg gtacaagttc atggacgtgt ggggccaggg cacactggtg 360
accgtcagct cagctagcac caagggcccc agcgtgttcc ccctggcccc cagcagcaag 420
agcaccagcg gcggcacagc cgccctgggc tgcctggtga aggactactt ccccgagccc 480
gtgaccgtgt cctggaacag cggagccctg acctccggcg tgcacacctt ccccgccgtg 540
ctgcagagca gcggcctgta cagcctgtcc agcgtggtga cagtgcccag cagcagcctg 600
ggcacccaga cctacatctg caacgtgaac cacaagccca gcaacaccaa ggtggacaag 660
agagtggagc ccaagagctg cgacaagacc cacacctgcc ccccctgccc agccccagag 720
gcagcgggcg gaccctccgt gttcctgttc ccccccaagc ccaaggacac cctgatgatc 780
agcaggaccc ccgaggtgac ctgcgtggtg gtggacgtga gccacgagga cccagaggtg 840
aagttcaact ggtacgtgga cggcgtggag gtgcacaacg ccaagaccaa gcccagagag 900
gagcagtaca acagcaccta cagggtggtg tccgtgctga ccgtgctgca ccaggactgg 960
ctgaacggca aggaatacaa gtgcaaggtc tccaacaagg ccctgccagc ccccatcgaa 1020
aagaccatca gcaaggccaa gggccagcca cgggagcccc aggtgtacac cctgcccccc 1080
tcccgggagg agatgaccaa gaaccaggtg tccctgacct gtctggtgaa gggcttctac 1140
cccagcgaca tcgccgtgga gtgggagagc aacggccagc ccgagaacaa ctacaagacc 1200
acccccccag tgctggacag cgacggcagc ttcttcctgt acagcaagct gaccgtggac 1260
aagtccaggt ggcagcaggg caacgtgttc agctgcagcg tgatgcacga agcgctgcac 1320
aaccactaca cccagaagag cctgagcctg tcccccggca agggcggctc cggcggaagc 1380
gatatcgtgc tgacacagag ccctgccacc ctgtctctga gccctggcga gagagccacc 1440
ctgagctgcc gggccagcca gttcatcggc tcccgctacc tggcctggta tcagcagaag 1500
cccggacagg ctcccagact gctgatctac ggcgccagca acagagctac cggcgtgccc 1560
gccagatttt ctggcagcgg cagcggcacc gacttcaccc tgaccatcag cagcctggaa 1620
cccgaggact tcgccaccta ctactgccag cagtactacg actaccccca gaccttcggc 1680
cagggcacca aggtggagat caagggcgga ggcggatccg ggggtggcgg aagtggaggc 1740
ggaggaagcg gagggggcgg aagccaggtg caattgaaag agtccggccc tgccctggtg 1800
aagcctaccc agaccctgac cctgacatgc accttcagcg gcttcagcct gagcaacaga 1860
ggcggcggag tgggctggat cagacagcct cccggcaagg ccctggaatg gctggcctgg 1920
atcgactggg acgacgacaa gagctacagc accagcctga aaacccggct gaccatctcc 1980
aaggacacca gcaagaacca ggtggtgctc accatgacca acatggacgc cgtggacacc 2040
gccacctatt attgcgcccg gatgcatctg cccctggtgt tcgatagctg gggccaggga 2100
accctggtga cagtgtccag c 2121
<210> 175
<211> 707
<212> PRT
<213> Homo Sapien
<400> 175
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Val Ile Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Ser Gly Val Asn Thr His Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Gly Ala Thr Ala Asn Asn Ile Arg Tyr Lys Phe Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
115 120 125
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
130 135 140
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
145 150 155 160
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
165 170 175
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
180 185 190
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
195 200 205
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro
210 215 220
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
225 230 235 240
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
290 295 300
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
305 310 315 320
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
325 330 335
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
340 345 350
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
355 360 365
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
370 375 380
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
385 390 395 400
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
405 410 415
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
420 425 430
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
435 440 445
Ser Leu Ser Pro Gly Lys Gly Gly Ser Gly Gly Ser Asp Ile Val Leu
450 455 460
Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr
465 470 475 480
Leu Ser Cys Arg Ala Ser Gln Phe Ile Gly Ser Arg Tyr Leu Ala Trp
485 490 495
Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Gly Ala
500 505 510
Ser Asn Arg Ala Thr Gly Val Pro Ala Arg Phe Ser Gly Ser Gly Ser
515 520 525
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu Asp Phe
530 535 540
Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Tyr Pro Gln Thr Phe Gly
545 550 555 560
Gln Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly
565 570 575
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu
580 585 590
Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln Thr Leu Thr Leu
595 600 605
Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Asn Arg Gly Gly Gly Val
610 615 620
Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu Trp Leu Ala Trp
625 630 635 640
Ile Asp Trp Asp Asp Asp Lys Ser Tyr Ser Thr Ser Leu Lys Thr Arg
645 650 655
Leu Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Val Val Leu Thr Met
660 665 670
Thr Asn Met Thr Ala Val Asp Thr Ala Thr Tyr Tyr Cys Ala Arg Met
675 680 685
His Leu Pro Leu Val Phe Asp Ser Trp Gly Gln Gly Thr Leu Val Thr
690 695 700
Val Ser Ser
705
<210> 176
<211> 2121
<212> DNA
<213> Homo Sapien
<400> 176
caggtgcaat tggtcgagtc tggcggagga ctggtgcagc ctggtggcag cctgagactg 60
agctgcgccg ccagcggctt caccttcagc gactacgtga tcaactgggt gcgacaggcc 120
cctggaaagg gcctggaatg ggtgtccggc atctcttggt ctggcgtgaa cacccactac 180
gccgacagcg tgaagggccg gttcaccatc agccgggaca acagcaagaa caccctgtac 240
ctgcagatga acagcctgag agccgaggac accgccgtgt actactgtgc cagactgggc 300
gccaccgcca acaacatccg gtacaagttc atggacgtgt ggggccaggg cacactggtg 360
accgtcagct cagctagcac caagggcccc agcgtgttcc ccctggcccc cagcagcaag 420
agcaccagcg gcggcacagc cgccctgggc tgcctggtga aggactactt ccccgagccc 480
gtgaccgtgt cctggaacag cggagccctg acctccggcg tgcacacctt ccccgccgtg 540
ctgcagagca gcggcctgta cagcctgtcc agcgtggtga cagtgcccag cagcagcctg 600
ggcacccaga cctacatctg caacgtgaac cacaagccca gcaacaccaa ggtggacaag 660
agagtggagc ccaagagctg cgacaagacc cacacctgcc ccccctgccc agccccagag 720
gcagcgggcg gaccctccgt gttcctgttc ccccccaagc ccaaggacac cctgatgatc 780
agcaggaccc ccgaggtgac ctgcgtggtg gtggacgtga gccacgagga cccagaggtg 840
aagttcaact ggtacgtgga cggcgtggag gtgcacaacg ccaagaccaa gcccagagag 900
gagcagtaca acagcaccta cagggtggtg tccgtgctga ccgtgctgca ccaggactgg 960
ctgaacggca aggaatacaa gtgcaaggtc tccaacaagg ccctgccagc ccccatcgaa 1020
aagaccatca gcaaggccaa gggccagcca cgggagcccc aggtgtacac cctgcccccc 1080
tcccgggagg agatgaccaa gaaccaggtg tccctgacct gtctggtgaa gggcttctac 1140
cccagcgaca tcgccgtgga gtgggagagc aacggccagc ccgagaacaa ctacaagacc 1200
acccccccag tgctggacag cgacggcagc ttcttcctgt acagcaagct gaccgtggac 1260
aagtccaggt ggcagcaggg caacgtgttc agctgcagcg tgatgcacga agcgctgcac 1320
aaccactaca cccagaagag cctgagcctg tcccccggca agggcggctc cggcggaagc 1380
gatatcgtgc tgacacagag ccctgccacc ctgtctctga gccctggcga gagagccacc 1440
ctgagctgcc gggccagcca gttcatcggc tcccgctacc tggcctggta tcagcagaag 1500
cccggacagg ctcccagact gctgatctac ggcgccagca acagagctac cggcgtgccc 1560
gccagatttt ctggcagcgg cagcggcacc gacttcaccc tgaccatcag cagcctggaa 1620
cccgaggact tcgccaccta ctactgccag cagtactacg actaccccca gaccttcggc 1680
cagggcacca aggtggagat caagggcgga ggcggatccg ggggtggcgg aagtggaggc 1740
ggaggaagcg gagggggcgg aagccaggtg caattgaaag agtccggccc tgccctggtg 1800
aagcctaccc agaccctgac cctgacatgc accttcagcg gcttcagcct gagcaacaga 1860
ggcggcggag tgggctggat cagacagcct cccggcaagg ccctggaatg gctggcctgg 1920
atcgactggg acgacgacaa gagctacagc accagcctga aaacccggct gaccatctcc 1980
aaggacacca gcaagaacca ggtggtgctc accatgacca acatgaccgc cgtggacacc 2040
gccacctatt attgcgcccg gatgcatctg cccctggtgt tcgatagctg gggccaggga 2100
accctggtga cagtgtccag c 2121
<210> 177
<211> 464
<212> PRT
<213> Homo Sapien
<400> 177
Asp Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Phe Ile Gly Ser Arg
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Asn Arg Ala Thr Gly Val Pro Ala Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Tyr Pro
85 90 95
Gln Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly
100 105 110
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gln Val Gln Leu Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln
130 135 140
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Asn Arg
145 150 155 160
Gly Gly Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
165 170 175
Trp Leu Ala Trp Ile Asp Trp Asp Asp Asp Lys Ser Tyr Ser Thr Ser
180 185 190
Leu Lys Thr Arg Leu Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Val
195 200 205
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
210 215 220
Cys Ala Arg Met His Leu Pro Leu Val Phe Asp Ser Trp Gly Gln Gly
225 230 235 240
Thr Leu Val Thr Val Ser Ser Gly Gly Ser Gly Gly Ser Asp Ile Glu
245 250 255
Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln Thr Ala Arg
260 265 270
Ile Ser Cys Ser Gly Asp Ser Leu Arg Asn Lys Val Tyr Trp Tyr Gln
275 280 285
Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr Lys Asn Asn Arg
290 295 300
Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser Asn Ser Gly Asn Thr
305 310 315 320
Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu Asp Glu Ala Asp Tyr
325 330 335
Tyr Cys Gln Ser Tyr Asp Gly Gln Lys Ser Leu Val Phe Gly Gly Gly
340 345 350
Thr Lys Leu Thr Val Leu Gly Gln Pro Lys Ala Ala Pro Ser Val Thr
355 360 365
Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu
370 375 380
Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val Thr Val Ala Trp
385 390 395 400
Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu Thr Thr Thr Pro
405 410 415
Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu
420 425 430
Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser Cys Gln Val Thr
435 440 445
His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
450 455 460
<210> 178
<211> 1392
<212> DNA
<213> Homo Sapien
<400> 178
gatatcgtgc tgacacagag ccctgccacc ctgtctctga gccctggcga gagagccacc 60
ctgagctgcc gggccagcca gttcatcggc tcccgctacc tggcctggta tcagcagaag 120
cccggacagg ctcccagact gctgatctac ggcgccagca acagagctac cggcgtgccc 180
gccagatttt ctggcagcgg cagcggcacc gacttcaccc tgaccatcag cagcctggaa 240
cccgaggact tcgccaccta ctactgccag cagtactacg actaccccca gaccttcggc 300
cagggcacca aggtggagat caagggcgga ggcggatccg ggggtggcgg aagtggaggc 360
ggaggaagcg gagggggcgg aagccaggtg caattgaaag agtccggccc tgccctggtg 420
aagcctaccc agaccctgac cctgacatgc accttcagcg gcttcagcct gagcaacaga 480
ggcggcggag tgggctggat cagacagcct cccggcaagg ccctggaatg gctggcctgg 540
atcgactggg acgacgacaa gagctacagc accagcctga aaacccggct gaccatctcc 600
aaggacacca gcaagaacca ggtggtgctc accatgacca acatggaccc cgtggacacc 660
gccacctatt attgcgcccg gatgcatctg cccctggtgt tcgatagctg gggccaggga 720
accctggtga cagtgtccag cggcggctcc ggcggaagcg acatcgagct gacccagccc 780
ccttctgtgt ctgtggcgcc cgggcagacc gccagaatca gctgcagcgg cgacagcctg 840
cggaacaagg tgtactggta tcagcagaag cccggccagg ctcccgtgct ggtgatctac 900
aagaacaacc ggcccagcgg catccctgag cggttcagcg gcagcaacag cggcaatacc 960
gccaccctga ccatcagcgg cacccaggcc gaagatgagg ccgactacta ctgccagagc 1020
tacgacggcc agaaaagcct ggtgttcggc ggaggcacca agcttaccgt gctgggccag 1080
cccaaagccg cccctagcgt gaccctgttc ccccccagca gcgaggaact gcaggccaac 1140
aaggccaccc tggtctgcct gatcagcgac ttctaccctg gcgccgtgac cgtggcctgg 1200
aaggccgaca gcagccccgt gaaggccggc gtggagacaa ccacccccag caagcagagc 1260
aacaacaagt acgccgccag cagctacctg agcctgaccc ccgagcagtg gaagagccac 1320
agaagctaca gctgccaggt cacccacgag ggcagcaccg tggagaaaac cgtggccccc 1380
accgagtgca gc 1392
<210> 179
<211> 124
<212> PRT
<213> Homo Sapien
<400> 179
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Val Ile Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Ser Gly Val Asn Thr His Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Gly Ala Thr Ala Asn Asn Ile Arg Tyr Lys Phe Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 180
<211> 372
<212> DNA
<213> Homo Sapien
<400> 180
caggtgcaat tggtcgagtc tggcggagga ctggtgcagc ctggtggcag cctgagactg 60
agctgcgccg ccagcggctt caccttcagc gactacgtga tcaactgggt gcgacaggcc 120
cctggaaagg gcctggaatg ggtgtccggc atctcttggt ctggcgtgaa cacccactac 180
gccgacagcg tgaagggccg gttcaccatc agccgggaca acagcaagaa caccctgtac 240
ctgcagatga acagcctgag agccgaggac accgccgtgt actactgtgc cagactgggc 300
gccaccgcca acaacatccg gtacaagttc atggacgtgt ggggccaggg cacactggtg 360
accgtcagct ca 372
<210> 181
<211> 454
<212> PRT
<213> Homo Sapien
<400> 181
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Val Ile Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Ser Gly Val Asn Thr His Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Gly Ala Thr Ala Asn Asn Ile Arg Tyr Lys Phe Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
115 120 125
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
130 135 140
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
145 150 155 160
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
165 170 175
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
180 185 190
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
195 200 205
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro
210 215 220
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
225 230 235 240
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
290 295 300
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
305 310 315 320
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
325 330 335
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
340 345 350
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
355 360 365
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
370 375 380
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
385 390 395 400
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
405 410 415
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
420 425 430
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
435 440 445
Ser Leu Ser Pro Gly Lys
450
<210> 182
<211> 1362
<212> DNA
<213> Homo Sapien
<400> 182
caggtgcaat tggtcgagtc tggcggagga ctggtgcagc ctggtggcag cctgagactg 60
agctgcgccg ccagcggctt caccttcagc gactacgtga tcaactgggt gcgacaggcc 120
cctggaaagg gcctggaatg ggtgtccggc atctcttggt ctggcgtgaa cacccactac 180
gccgacagcg tgaagggccg gttcaccatc agccgggaca acagcaagaa caccctgtac 240
ctgcagatga acagcctgag agccgaggac accgccgtgt actactgtgc cagactgggc 300
gccaccgcca acaacatccg gtacaagttc atggacgtgt ggggccaggg cacactggtg 360
accgtcagct cagctagcac caagggcccc agcgtgttcc ccctggcccc cagcagcaag 420
agcaccagcg gcggcacagc cgccctgggc tgcctggtga aggactactt ccccgagccc 480
gtgaccgtgt cctggaacag cggagccctg acctccggcg tgcacacctt ccccgccgtg 540
ctgcagagca gcggcctgta cagcctgtcc agcgtggtga cagtgcccag cagcagcctg 600
ggcacccaga cctacatctg caacgtgaac cacaagccca gcaacaccaa ggtggacaag 660
agagtggagc ccaagagctg cgacaagacc cacacctgcc ccccctgccc agccccagag 720
gcagcgggcg gaccctccgt gttcctgttc ccccccaagc ccaaggacac cctgatgatc 780
agcaggaccc ccgaggtgac ctgcgtggtg gtggacgtga gccacgagga cccagaggtg 840
aagttcaact ggtacgtgga cggcgtggag gtgcacaacg ccaagaccaa gcccagagag 900
gagcagtaca acagcaccta cagggtggtg tccgtgctga ccgtgctgca ccaggactgg 960
ctgaacggca aggaatacaa gtgcaaggtc tccaacaagg ccctgccagc ccccatcgaa 1020
aagaccatca gcaaggccaa gggccagcca cgggagcccc aggtgtacac cctgcccccc 1080
tcccgggagg agatgaccaa gaaccaggtg tccctgacct gtctggtgaa gggcttctac 1140
cccagcgaca tcgccgtgga gtgggagagc aacggccagc ccgagaacaa ctacaagacc 1200
acccccccag tgctggacag cgacggcagc ttcttcctgt acagcaagct gaccgtggac 1260
aagtccaggt ggcagcaggg caacgtgttc agctgcagcg tgatgcacga ggccctgcac 1320
aaccactaca cccagaagag cctgagcctg tcccccggca ag 1362
<210> 183
<211> 108
<212> PRT
<213> Homo Sapien
<400> 183
Asp Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Phe Ile Gly Ser Arg
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Asn Arg Ala Thr Gly Val Pro Ala Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Tyr Pro
85 90 95
Gln Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 184
<211> 324
<212> DNA
<213> Homo Sapien
<400> 184
gatatcgtgc tgacccagag cccggcgacc ctgagcctgt ctccgggcga acgtgcgacc 60
ctgagctgca gagcgagcca gtttattggt tctcgttatc tggcttggta ccagcagaaa 120
ccaggtcaag caccgcgtct attaatttat ggtgcttcta atcgtgcaac tggggtcccg 180
gcgcgtttta gcggctctgg atccggcacg gattttaccc tgaccattag cagcctggaa 240
cctgaagact ttgcgactta ttattgccag cagtattatg attatcctca gacctttggc 300
cagggtacga aagttgaaat taaa 324
<210> 185
<211> 215
<212> PRT
<213> Homo Sapien
<400> 185
Asp Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Phe Ile Gly Ser Arg
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Asn Arg Ala Thr Gly Val Pro Ala Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Tyr Pro
85 90 95
Gln Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala
100 105 110
Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser
115 120 125
Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu
130 135 140
Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser
145 150 155 160
Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu
165 170 175
Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val
180 185 190
Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys
195 200 205
Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 186
<211> 645
<212> DNA
<213> Homo Sapien
<400> 186
gatatcgtgc tgacccagag cccggcgacc ctgagcctgt ctccgggcga acgtgcgacc 60
ctgagctgca gagcgagcca gtttattggt tctcgttatc tggcttggta ccagcagaaa 120
ccaggtcaag caccgcgtct attaatttat ggtgcttcta atcgtgcaac tggggtcccg 180
gcgcgtttta gcggctctgg atccggcacg gattttaccc tgaccattag cagcctggaa 240
cctgaagact ttgcgactta ttattgccag cagtattatg attatcctca gacctttggc 300
cagggtacga aagttgaaat taaacgtacg gtggccgctc ccagcgtgtt catcttcccc 360
cccagcgacg agcagctgaa gagcggcacc gccagcgtgg tgtgcctgct gaacaacttc 420
tacccccggg aggccaaggt gcagtggaag gtggacaacg ccctgcagag cggcaacagc 480
caggagagcg tcaccgagca ggacagcaag gactccacct acagcctgag cagcaccctg 540
accctgagca aggccgacta cgagaagcat aaggtgtacg cctgcgaggt gacccaccag 600
ggcctgtcca gccccgtgac caagagcttc aacaggggcg agtgc 645
<210> 187
<211> 699
<212> PRT
<213> Homo Sapien
<400> 187
Gln Val Gln Leu Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Asn Arg
20 25 30
Gly Gly Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala Trp Ile Asp Trp Asp Asp Asp Lys Ser Tyr Ser Thr Ser
50 55 60
Leu Lys Thr Arg Leu Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Met His Leu Pro Leu Val Phe Asp Ser Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys
210 215 220
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro
225 230 235 240
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
245 250 255
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
260 265 270
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
275 280 285
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
290 295 300
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
305 310 315 320
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
325 330 335
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
340 345 350
Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr
355 360 365
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
370 375 380
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
385 390 395 400
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
405 410 415
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
420 425 430
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
435 440 445
Lys Gly Gly Ser Gly Gly Ser Gln Val Gln Leu Val Glu Ser Gly Gly
450 455 460
Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser
465 470 475 480
Gly Phe Thr Phe Ser Asp Tyr Val Ile Asn Trp Val Arg Gln Ala Pro
485 490 495
Gly Lys Gly Leu Glu Trp Val Ser Gly Ile Ser Trp Ser Gly Val Asn
500 505 510
Thr His Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
515 520 525
Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu
530 535 540
Asp Thr Ala Val Tyr Tyr Cys Ala Arg Leu Gly Ala Thr Ala Asn Asn
545 550 555 560
Ile Arg Tyr Lys Phe Met Asp Val Trp Gly Gln Gly Thr Leu Val Thr
565 570 575
Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
580 585 590
Gly Ser Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro
595 600 605
Gly Gln Thr Ala Arg Ile Ser Cys Ser Gly Asp Ser Leu Arg Asn Lys
610 615 620
Val Tyr Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile
625 630 635 640
Tyr Lys Asn Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
645 650 655
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu
660 665 670
Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Gly Gln Lys Ser Leu
675 680 685
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
690 695
<210> 188
<211> 2097
<212> DNA
<213> Homo Sapien
<400> 188
caggtgcaat tgaaagaaag cggcccggcc ctggtgaaac cgacccaaac cctgaccctg 60
acctgtacct tttccggatt tagcctgtct aatcgtggtg gtggtgtggg ttggattcgc 120
cagccgcctg ggaaagccct cgagtggctg gcttggatcg attgggatga tgataagtct 180
tatagcacca gcctgaaaac gcgtctgacc attagcaaag atacttcgaa aaatcaggtg 240
gtgctgacta tgaccaacat ggacccggtg gatacggcca cctattattg cgcgcgtatg 300
catcttcctc ttgtttttga ttcttggggc caaggcaccc tggtgacggt tagctcagct 360
agcaccaagg gccccagcgt gttccccctg gcccccagca gcaagagcac cagcggcggc 420
acagccgccc tgggctgcct ggtgaaggac tacttccccg agcccgtgac cgtgtcctgg 480
aacagcggag ccctgacctc cggcgtgcac accttccccg ccgtgctgca gagcagcggc 540
ctgtacagcc tgtccagcgt ggtgacagtg cccagcagca gcctgggcac ccagacctac 600
atctgcaacg tgaaccacaa gcccagcaac accaaggtgg acaagagagt ggagcccaag 660
agctgcgaca agacccacac ctgccccccc tgcccagccc cagaggcagc gggcggaccc 720
tccgtgttcc tgttcccccc caagcccaag gacaccctga tgatcagcag gacccccgag 780
gtgacctgcg tggtggtgga cgtgagccac gaggacccag aggtgaagtt caactggtac 840
gtggacggcg tggaggtgca caacgccaag accaagccca gagaggagca gtacaacagc 900
acctacaggg tggtgtccgt gctgaccgtg ctgcaccagg actggctgaa cggcaaggaa 960
tacaagtgca aggtctccaa caaggccctg ccagccccca tcgaaaagac catcagcaag 1020
gccaagggcc agccacggga gccccaggtg tacaccctgc ccccctcccg ggaggagatg 1080
accaagaacc aggtgtccct gacctgtctg gtgaagggct tctaccccag cgacatcgcc 1140
gtggagtggg agagcaacgg ccagcccgag aacaactaca agaccacccc cccagtgctg 1200
gacagcgacg gcagcttctt cctgtacagc aagctgaccg tggacaagtc caggtggcag 1260
cagggcaacg tgttcagctg cagcgtgatg cacgaagcgc tgcacaacca ctacacccag 1320
aagagcctga gcctgtcccc cggcaagggc ggctccggcg gaagccaggt tcaattggtt 1380
gaaagcggtg gtggtctggt tcagcctggt ggtagcctgc gtctgagctg tgcagcaagc 1440
ggttttacct ttagcgatta tgtgattaat tgggttcgtc aggcaccggg taaaggtctg 1500
gaatgggtta gcggtattag ctggtcaggt gttaataccc attatgcaga tagcgtgaaa 1560
ggtcgtttta ccattagccg tgataatagc aaaaataccc tgtatctgca gatgaatagc 1620
ctgcgtgcag aagataccgc agtttattat tgtgcacgtc tgggtgcaac cgcaaataat 1680
attcgctata aatttatgga tgtgtggggt cagggtacac tagttaccgt tagcagtggt 1740
ggtggtggta gcggtggtgg cggatctggt ggcggtggca gtgatatcga actgacccag 1800
cctccgagcg ttagcgttgc accgggtcag accgcacgta ttagctgtag cggtgatagt 1860
ctgcgtaata aagtttattg gtatcagcag aaaccgggtc aggctccggt tctggttatt 1920
tataaaaata atcgtccgag cggtattccg gaacgtttta gcggtagcaa tagcggtaat 1980
accgcaaccc tgaccattag cggcacccag gcagaagatg aagccgatta ttattgtcag 2040
agctatgatg gtcagaaaag cctggttttt ggtggtggca ccaagcttac cgttctg 2097
<210> 189
<211> 704
<212> PRT
<213> Homo Sapien
<400> 189
Gln Val Gln Leu Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Asn Arg
20 25 30
Gly Gly Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala Trp Ile Asp Trp Asp Asp Asp Lys Ser Tyr Ser Thr Ser
50 55 60
Leu Lys Thr Arg Leu Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Met His Leu Pro Leu Val Phe Asp Ser Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys
210 215 220
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro
225 230 235 240
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
245 250 255
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
260 265 270
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
275 280 285
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
290 295 300
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
305 310 315 320
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
325 330 335
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
340 345 350
Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr
355 360 365
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
370 375 380
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
385 390 395 400
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
405 410 415
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
420 425 430
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
435 440 445
Lys Gly Gly Ser Gly Gly Ser Gln Val Gln Leu Val Glu Ser Gly Gly
450 455 460
Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser
465 470 475 480
Gly Phe Thr Phe Ser Asp Tyr Val Ile Asn Trp Val Arg Gln Ala Pro
485 490 495
Gly Lys Gly Leu Glu Trp Val Ser Gly Ile Ser Trp Ser Gly Val Asn
500 505 510
Thr His Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
515 520 525
Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu
530 535 540
Asp Thr Ala Val Tyr Tyr Cys Ala Arg Leu Gly Ala Thr Ala Asn Asn
545 550 555 560
Ile Arg Tyr Lys Phe Met Asp Val Trp Gly Gln Gly Thr Leu Val Thr
565 570 575
Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
580 585 590
Gly Ser Gly Gly Gly Gly Ser Asp Ile Glu Leu Thr Gln Pro Pro Ser
595 600 605
Val Ser Val Ala Pro Gly Gln Thr Ala Arg Ile Ser Cys Ser Gly Asp
610 615 620
Ser Leu Arg Asn Lys Val Tyr Trp Tyr Gln Gln Lys Pro Gly Gln Ala
625 630 635 640
Pro Val Leu Val Ile Tyr Lys Asn Asn Arg Pro Ser Gly Ile Pro Glu
645 650 655
Arg Phe Ser Gly Ser Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser
660 665 670
Gly Thr Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp
675 680 685
Gly Gln Lys Ser Leu Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
690 695 700
<210> 190
<211> 2112
<212> DNA
<213> Homo Sapien
<400> 190
caggtgcaat tgaaagaaag cggcccggcc ctggtgaaac cgacccaaac cctgaccctg 60
acctgtacct tttccggatt tagcctgtct aatcgtggtg gtggtgtggg ttggattcgc 120
cagccgcctg ggaaagccct cgagtggctg gcttggatcg attgggatga tgataagtct 180
tatagcacca gcctgaaaac gcgtctgacc attagcaaag atacttcgaa aaatcaggtg 240
gtgctgacta tgaccaacat ggacccggtg gatacggcca cctattattg cgcgcgtatg 300
catcttcctc ttgtttttga ttcttggggc caaggcaccc tggtgacggt tagctcagct 360
agcaccaagg gccccagcgt gttccccctg gcccccagca gcaagagcac cagcggcggc 420
acagccgccc tgggctgcct ggtgaaggac tacttccccg agcccgtgac cgtgtcctgg 480
aacagcggag ccctgacctc cggcgtgcac accttccccg ccgtgctgca gagcagcggc 540
ctgtacagcc tgtccagcgt ggtgacagtg cccagcagca gcctgggcac ccagacctac 600
atctgcaacg tgaaccacaa gcccagcaac accaaggtgg acaagagagt ggagcccaag 660
agctgcgaca agacccacac ctgccccccc tgcccagccc cagaggcagc gggcggaccc 720
tccgtgttcc tgttcccccc caagcccaag gacaccctga tgatcagcag gacccccgag 780
gtgacctgcg tggtggtgga cgtgagccac gaggacccag aggtgaagtt caactggtac 840
gtggacggcg tggaggtgca caacgccaag accaagccca gagaggagca gtacaacagc 900
acctacaggg tggtgtccgt gctgaccgtg ctgcaccagg actggctgaa cggcaaggaa 960
tacaagtgca aggtctccaa caaggccctg ccagccccca tcgaaaagac catcagcaag 1020
gccaagggcc agccacggga gccccaggtg tacaccctgc ccccctcccg ggaggagatg 1080
accaagaacc aggtgtccct gacctgtctg gtgaagggct tctaccccag cgacatcgcc 1140
gtggagtggg agagcaacgg ccagcccgag aacaactaca agaccacccc cccagtgctg 1200
gacagcgacg gcagcttctt cctgtacagc aagctgaccg tggacaagtc caggtggcag 1260
cagggcaacg tgttcagctg cagcgtgatg cacgaagcgc tgcacaacca ctacacccag 1320
aagagcctga gcctgtcccc cggcaagggc ggctccggcg gaagccaggt tcaattggtt 1380
gaaagcggtg gtggtctggt tcagcctggt ggtagcctgc gtctgagctg tgcagcaagc 1440
ggttttacct ttagcgatta tgtgattaat tgggttcgtc aggcaccggg taaaggtctg 1500
gaatgggtta gcggtattag ctggtcaggt gttaataccc attatgcaga tagcgtgaaa 1560
ggtcgtttta ccattagccg tgataatagc aaaaataccc tgtatctgca gatgaatagc 1620
ctgcgtgcag aagataccgc agtttattat tgtgcacgtc tgggtgcaac cgcaaataat 1680
attcgctata aatttatgga tgtgtggggt cagggtacac tagttaccgt tagcagtggt 1740
ggtggtggta gcggtggtgg cggatctggt ggcggtggtt caggtggtgg tggcagtgat 1800
atcgaactga cccagcctcc gagcgttagc gttgcaccgg gtcagaccgc acgtattagc 1860
tgtagcggtg atagtctgcg taataaagtt tattggtatc agcagaaacc gggtcaggct 1920
ccggttctgg ttatttataa aaataatcgt ccgagcggta ttccggaacg ttttagcggt 1980
agcaatagcg gtaataccgc aaccctgacc attagcggca cccaggcaga agatgaagcc 2040
gattattatt gtcagagcta tgatggtcag aaaagcctgg tttttggtgg tggcaccaag 2100
cttaccgttc tg 2112
<210> 191
<211> 105
<212> PRT
<213> Homo Sapien
<400> 191
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Ser Cys Ser Gly Asp Ser Leu Arg Asn Lys Val Tyr
20 25 30
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr Lys
35 40 45
Asn Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser Asn Ser
50 55 60
Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu Asp Glu
65 70 75 80
Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Gly Gln Lys Ser Leu Val Phe
85 90 95
Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 192
<211> 315
<212> DNA
<213> Homo Sapien
<400> 192
gacatcgagc tgactcagcc ccctagcgtg tcagtggctc ctggccagac cgctagaatt 60
agctgtagcg gcgatagcct gcgtaacaag gtctactggt atcagcagaa gcccggccag 120
gcccctgtgc tggtcatcta taagaacaat aggcctagcg gcatccccga gcggtttagc 180
ggctctaata gcggcaacac cgctaccctg actattagcg gcactcaggc cgaggacgag 240
gccgactact actgtcagtc ctacgacggc cagaagtcac tggtctttgg cggcggaact 300
aagctgaccg tgctg 315
<210> 193
<211> 211
<212> PRT
<213> Homo Sapien
<400> 193
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Ser Cys Ser Gly Asp Ser Leu Arg Asn Lys Val Tyr
20 25 30
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr Lys
35 40 45
Asn Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser Asn Ser
50 55 60
Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu Asp Glu
65 70 75 80
Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Gly Gln Lys Ser Leu Val Phe
85 90 95
Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln Pro Lys Ala Ala Pro
100 105 110
Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn Lys
115 120 125
Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val Thr
130 135 140
Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu Thr
145 150 155 160
Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser Tyr
165 170 175
Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser Cys
180 185 190
Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro Thr
195 200 205
Glu Cys Ser
210
<210> 194
<211> 633
<212> DNA
<213> Homo Sapien
<400> 194
gacatcgagc tgactcagcc ccctagcgtg tcagtggctc ctggccagac cgctagaatt 60
agctgtagcg gcgatagcct gcgtaacaag gtctactggt atcagcagaa gcccggccag 120
gcccctgtgc tggtcatcta taagaacaat aggcctagcg gcatccccga gcggtttagc 180
ggctctaata gcggcaacac cgctaccctg actattagcg gcactcaggc cgaggacgag 240
gccgactact actgtcagtc ctacgacggc cagaagtcac tggtctttgg cggcggaact 300
aagctgaccg tgctgggaca gcctaaggct gcccccagcg tgaccctgtt cccccccagc 360
agcgaggagc tgcaggccaa caaggccacc ctggtgtgcc tgatcagcga cttctaccca 420
ggcgccgtga ccgtggcctg gaaggccgac agcagccccg tgaaggccgg cgtggagacc 480
accaccccca gcaagcagag caacaacaag tacgccgcca gcagctacct gagcctgacc 540
cccgagcagt ggaagagcca caggtcctac agctgccagg tgacccacga gggcagcacc 600
gtggaaaaga ccgtggcccc aaccgagtgc agc 633
<210> 195
<211> 707
<212> PRT
<213> Homo Sapien
<400> 195
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Val Ile Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Ser Gly Val Asn Thr His Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Gly Ala Thr Ala Asn Asn Ile Arg Tyr Lys Phe Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
115 120 125
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
130 135 140
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
145 150 155 160
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
165 170 175
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
180 185 190
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
195 200 205
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro
210 215 220
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
225 230 235 240
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
290 295 300
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
305 310 315 320
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
325 330 335
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
340 345 350
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
355 360 365
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
370 375 380
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
385 390 395 400
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
405 410 415
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
420 425 430
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
435 440 445
Ser Leu Ser Pro Gly Lys Gly Gly Ser Gly Gly Ser Asp Ile Val Leu
450 455 460
Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr
465 470 475 480
Leu Ser Cys Arg Ala Ser Gln Phe Ile Gly Ser Arg Tyr Leu Ala Trp
485 490 495
Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Gly Ala
500 505 510
Ser Asn Arg Ala Thr Gly Val Pro Ala Arg Phe Ser Gly Ser Gly Ser
515 520 525
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu Asp Phe
530 535 540
Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Tyr Pro Gln Thr Phe Gly
545 550 555 560
Gln Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly
565 570 575
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu
580 585 590
Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln Thr Leu Thr Leu
595 600 605
Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Asn Arg Gly Gly Gly Val
610 615 620
Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu Trp Leu Ala Trp
625 630 635 640
Ile Asp Trp Asp Asp Asp Lys Ser Tyr Ser Thr Ser Leu Lys Thr Arg
645 650 655
Leu Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Val Val Leu Thr Met
660 665 670
Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr Cys Ala Arg Met
675 680 685
His Leu Pro Leu Val Phe Asp Ser Trp Gly Gln Gly Thr Leu Val Thr
690 695 700
Val Ser Ser
705
<210> 196
<211> 2121
<212> DNA
<213> Homo Sapien
<400> 196
caggtgcagc tggtggaatc aggcggagga ctggtccagc ctggcggatc acttagactg 60
agctgtgccg ctagtggctt cacctttagc gactatgtga ttaactgggt ccgacaggcc 120
cctggcaagg gactggaatg ggtgtcaggc attagttgga gcggcgtgaa cactcactac 180
gccgatagcg tgaagggccg gttcactatt agccgggata actctaagaa caccctgtac 240
ctgcagatga atagcctgag agccgaggac accgccgtct actactgcgc tagactgggc 300
gctaccgcta acaacatccg ctataagttc atggacgtgt ggggccaggg caccctggtc 360
acagtgtctt cagctagcac taagggcccc tcagtgttcc ccctggcccc tagctctaag 420
tctactagcg gtggcaccgc cgctctgggc tgcctggtca aggactactt ccccgagccc 480
gtgaccgtgt cttggaatag cggcgctctg actagcggag tgcacacctt ccccgccgtg 540
ctgcagtcta gcggcctgta tagcctgtct agcgtcgtga ccgtgcctag ctctagcctg 600
ggcactcaga cctatatctg taacgtgaac cacaagccta gtaacactaa ggtggacaag 660
cgggtggaac ctaagtcttg cgataagact cacacctgtc ccccctgccc tgccccagaa 720
gctgctggcg gacctagcgt gttcctgttc ccacctaagc ctaaagacac cctgatgatt 780
agtaggaccc ccgaagtgac ctgcgtggtg gtggacgtca gccacgagga ccctgaagtg 840
aagttcaatt ggtatgtgga cggcgtggaa gtgcacaacg ctaagactaa gcctagagag 900
gaacagtata actccaccta tagggtggtg tcagtgctga ccgtgctgca ccaggactgg 960
ctgaacggca aagagtataa gtgtaaagtc tctaacaagg ccctgcctgc ccctatcgaa 1020
aagactatct ctaaggctaa gggccagcct agagaacccc aggtctacac cctgccccct 1080
agtagagaag agatgactaa gaatcaggtg tccctgacct gtctggtcaa gggcttctac 1140
cctagcgata tcgccgtgga gtgggagtct aacggccagc ccgagaacaa ctataagact 1200
accccccctg tgctggatag cgacggctct ttcttcctgt actctaaact gaccgtggac 1260
aagtctaggt ggcagcaggg caacgtgttc agctgtagcg tgatgcacga ggccctgcac 1320
aatcactaca ctcagaagtc actgagcctg agtcccggca agggcggctc aggcggtagc 1380
gatatcgtgc tgactcagtc acccgctacc ctgagtctga gccctggcga gcgggctaca 1440
ctgagctgta gagctagtca gtttatcggc tcacgctacc tggcctggta tcagcagaag 1500
cccggccagg cccctagact gctgatctac ggcgctagta atagagctac cggcgtgccc 1560
gctaggttta gcggctcagg atcaggcacc gactttaccc tgactattag tagcctggaa 1620
cccgaggact tcgctaccta ctactgtcag cagtactacg actaccctca gaccttcggc 1680
cagggaacta aggtcgagat taagggcggt ggcggtagcg gcggaggcgg atcaggtggt 1740
ggtggtagtg gcggcggagg tagtcaggtc cagctgaaag agtcaggccc tgccctggtc 1800
aagcctactc agaccctgac cctgacctgc acttttagcg gctttagcct gagtaataga 1860
ggcggcggag tgggctggat tagacagcct ccaggcaaag ccctggagtg gctggcctgg 1920
atcgactggg acgacgataa gtcctactcc actagcctga aaactaggct gacaatcagc 1980
aaggacacta gtaaaaacca ggtggtgctg actatgacta atatggaccc cgtggacacc 2040
gctacctatt attgcgctag aatgcacctc ccactggtgt tcgatagctg gggtcaggga 2100
actctggtca cagtcagtag c 2121
<210> 197
<211> 105
<212> PRT
<213> Homo Sapien
<400> 197
Ser Tyr Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Ser Cys Ser Gly Asp Ser Leu Arg Asn Lys Val Tyr
20 25 30
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr Lys
35 40 45
Asn Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser Asn Ser
50 55 60
Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu Asp Glu
65 70 75 80
Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Gly Gln Lys Ser Leu Val Phe
85 90 95
Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 198
<211> 315
<212> DNA
<213> Homo Sapien
<400> 198
tcttacgagc tgacccagcc cccttccgtg tctgtggctc ctggccagac cgccagaatc 60
tcttgctccg gcgactccct gcggaacaag gtgtactggt atcagcagaa gcccggccag 120
gcccctgtgc tggtcatcta caagaacaac cggccctccg gcatccccga gagattctct 180
ggctccaact ccggcaacac cgccaccctg acaatctctg gcacacaggc cgaggacgag 240
gccgactact actgccagtc ctacgacggc cagaaatcac tggtgttcgg cggaggcacc 300
aagctgacag tgctg 315
<210> 199
<211> 211
<212> PRT
<213> Homo Sapien
<400> 199
Ser Tyr Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Ser Cys Ser Gly Asp Ser Leu Arg Asn Lys Val Tyr
20 25 30
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr Lys
35 40 45
Asn Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser Asn Ser
50 55 60
Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu Asp Glu
65 70 75 80
Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Gly Gln Lys Ser Leu Val Phe
85 90 95
Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln Pro Lys Ala Ala Pro
100 105 110
Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn Lys
115 120 125
Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val Thr
130 135 140
Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu Thr
145 150 155 160
Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser Tyr
165 170 175
Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser Cys
180 185 190
Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro Thr
195 200 205
Glu Cys Ser
210
<210> 200
<211> 633
<212> DNA
<213> Homo Sapien
<400> 200
tcttacgagc tgacccagcc cccttccgtg tctgtggctc ctggccagac cgccagaatc 60
tcttgctccg gcgactccct gcggaacaag gtgtactggt atcagcagaa gcccggccag 120
gcccctgtgc tggtcatcta caagaacaac cggccctccg gcatccccga gagattctct 180
ggctccaact ccggcaacac cgccaccctg acaatctctg gcacacaggc cgaggacgag 240
gccgactact actgccagtc ctacgacggc cagaaatcac tggtgttcgg cggaggcacc 300
aagctgacag tgctgggaca gcctaaggct gcccccagcg tgaccctgtt cccccccagc 360
agcgaggagc tgcaggccaa caaggccacc ctggtgtgcc tgatcagcga cttctaccca 420
ggcgccgtga ccgtggcctg gaaggccgac agcagccccg tgaaggccgg cgtggagacc 480
accaccccca gcaagcagag caacaacaag tacgccgcca gcagctacct gagcctgacc 540
cccgagcagt ggaagagcca caggtcctac agctgccagg tgacccacga gggcagcacc 600
gtggaaaaga ccgtggcccc aaccgagtgc agc 633
<210> 201
<211> 707
<212> PRT
<213> Homo Sapien
<400> 201
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Val Ile Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Ser Gly Val Asn Thr His Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Gly Ala Thr Ala Asn Asn Ile Arg Tyr Lys Phe Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
115 120 125
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
130 135 140
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
145 150 155 160
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
165 170 175
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
180 185 190
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
195 200 205
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro
210 215 220
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
225 230 235 240
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
290 295 300
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
305 310 315 320
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
325 330 335
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
340 345 350
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
355 360 365
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
370 375 380
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
385 390 395 400
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
405 410 415
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
420 425 430
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
435 440 445
Ser Leu Ser Pro Gly Lys Gly Gly Ser Gly Gly Ser Asp Ile Val Leu
450 455 460
Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr
465 470 475 480
Leu Ser Cys Arg Ala Ser Gln Phe Ile Gly Ser Arg Tyr Leu Ala Trp
485 490 495
Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Gly Ala
500 505 510
Ser Asn Arg Ala Thr Gly Val Pro Ala Arg Phe Ser Gly Ser Gly Ser
515 520 525
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu Asp Phe
530 535 540
Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Tyr Pro Gln Thr Phe Gly
545 550 555 560
Gln Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly
565 570 575
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu
580 585 590
Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln Thr Leu Thr Leu
595 600 605
Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Asn Arg Gly Gly Gly Val
610 615 620
Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu Trp Leu Ala Trp
625 630 635 640
Ile Asp Trp Asp Asp Asp Lys Ser Tyr Ser Thr Ser Leu Lys Thr Arg
645 650 655
Leu Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Val Val Leu Thr Met
660 665 670
Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr Cys Ala Arg Met
675 680 685
His Leu Pro Leu Val Phe Asp Ser Trp Gly Gln Gly Thr Leu Val Thr
690 695 700
Val Ser Ser
705
<210> 202
<211> 2121
<212> DNA
<213> Homo Sapien
<400> 202
caggtgcagc tggtggaatc aggcggagga ctggtccagc ctggcggatc acttagactg 60
agctgtgccg ctagtggctt cacctttagc gactatgtga ttaactgggt ccgacaggcc 120
cctggcaagg gactggaatg ggtgtcaggc attagttgga gcggcgtgaa cactcactac 180
gccgatagcg tgaagggccg gttcactatt agccgggata actctaagaa caccctgtac 240
ctgcagatga atagcctgag agccgaggac accgccgtct actactgcgc tagactgggc 300
gctaccgcta acaacatccg ctataagttc atggacgtgt ggggccaggg caccctggtc 360
acagtgtctt cagctagcac taagggcccc tcagtgttcc ccctggcccc tagctctaag 420
tctactagcg gtggcaccgc cgctctgggc tgcctggtca aggactactt ccccgagccc 480
gtgaccgtgt cttggaatag cggcgctctg actagcggag tgcacacctt ccccgccgtg 540
ctgcagtcta gcggcctgta tagcctgtct agcgtcgtga ccgtgcctag ctctagcctg 600
ggcactcaga cctatatctg taacgtgaac cacaagccta gtaacactaa ggtggacaag 660
cgggtggaac ctaagtcttg cgataagact cacacctgtc ccccctgccc tgccccagaa 720
gctgctggcg gacctagcgt gttcctgttc ccacctaagc ctaaagacac cctgatgatt 780
agtaggaccc ccgaagtgac ctgcgtggtg gtggacgtca gccacgagga ccctgaagtg 840
aagttcaatt ggtatgtgga cggcgtggaa gtgcacaacg ctaagactaa gcctagagag 900
gaacagtata actccaccta tagggtggtg tcagtgctga ccgtgctgca ccaggactgg 960
ctgaacggca aagagtataa gtgtaaagtc tctaacaagg ccctgcctgc ccctatcgaa 1020
aagactatct ctaaggctaa gggccagcct agagaacccc aggtctacac cctgccccct 1080
agtagagaag agatgactaa gaatcaggtg tccctgacct gtctggtcaa gggcttctac 1140
cctagcgata tcgccgtgga gtgggagtct aacggccagc ccgagaacaa ctataagact 1200
accccccctg tgctggatag cgacggctct ttcttcctgt actctaaact gaccgtggac 1260
aagtctaggt ggcagcaggg caacgtgttc agctgtagcg tgatgcacga ggccctgcac 1320
aatcactaca ctcagaagtc actgagcctg agtcccggca agggcggctc aggcggtagc 1380
gatatcgtgc tgactcagtc acccgctacc ctgagtctga gccctggcga gcgggctaca 1440
ctgagctgta gagctagtca gtttatcggc tcacgctacc tggcctggta tcagcagaag 1500
cccggccagg cccctagact gctgatctac ggcgctagta atagagctac cggcgtgccc 1560
gctaggttta gcggctcagg atcaggcacc gactttaccc tgactattag tagcctggaa 1620
cccgaggact tcgctaccta ctactgtcag cagtactacg actaccctca gaccttcggc 1680
cagggaacta aggtcgagat taagggcggt ggcggtagcg gcggaggcgg atcaggtggt 1740
ggtggtagtg gcggcggagg tagtcaggtc cagctgaaag agtcaggccc tgccctggtc 1800
aagcctactc agaccctgac cctgacctgc acttttagcg gctttagcct gagtaataga 1860
ggcggcggag tgggctggat tagacagcct ccaggcaaag ccctggagtg gctggcctgg 1920
atcgactggg acgacgataa gtcctactcc actagcctga aaactaggct gacaatcagc 1980
aaggacacta gtaaaaacca ggtggtgctg actatgacta atatggaccc cgtggacacc 2040
gctacctatt attgcgctag aatgcacctc ccactggtgt tcgatagctg gggtcaggga 2100
actctggtca cagtcagtag c 2121
<210> 203
<211> 105
<212> PRT
<213> Homo Sapien
<400> 203
Ser Tyr Glu Leu Thr Gln Pro Leu Ser Val Ser Val Ala Leu Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Ser Gly Asp Ser Leu Arg Asn Lys Val Tyr
20 25 30
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr Lys
35 40 45
Asn Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser Asn Ser
50 55 60
Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Ala Gln Ala Gly Asp Glu
65 70 75 80
Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Gly Gln Lys Ser Leu Val Phe
85 90 95
Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 204
<211> 315
<212> DNA
<213> Homo Sapien
<400> 204
agctacgagc tgactcagcc cctgagcgtg tcagtggctc tgggccagac cgctagaatc 60
acctgtagcg gcgatagcct gagaaacaag gtctactggt atcagcagaa gcccggccag 120
gcccctgtgc tggtcatcta taagaacaat aggcctagcg gcatccccga gcggtttagc 180
ggctctaata gcggcaacac cgctaccctg actattagta gggctcaggc cggcgacgag 240
gccgactact actgtcagtc ctacgacggc cagaagtcac tggtctttgg cggcggaact 300
aagctgaccg tgctg 315
<210> 205
<211> 211
<212> PRT
<213> Homo Sapien
<400> 205
Ser Tyr Glu Leu Thr Gln Pro Leu Ser Val Ser Val Ala Leu Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Ser Gly Asp Ser Leu Arg Asn Lys Val Tyr
20 25 30
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr Lys
35 40 45
Asn Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser Asn Ser
50 55 60
Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Ala Gln Ala Gly Asp Glu
65 70 75 80
Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Gly Gln Lys Ser Leu Val Phe
85 90 95
Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln Pro Lys Ala Ala Pro
100 105 110
Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn Lys
115 120 125
Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val Thr
130 135 140
Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu Thr
145 150 155 160
Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser Tyr
165 170 175
Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser Cys
180 185 190
Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro Thr
195 200 205
Glu Cys Ser
210
<210> 206
<211> 633
<212> DNA
<213> Homo Sapien
<400> 206
agctacgagc tgactcagcc cctgagcgtg tcagtggctc tgggccagac cgctagaatc 60
acctgtagcg gcgatagcct gagaaacaag gtctactggt atcagcagaa gcccggccag 120
gcccctgtgc tggtcatcta taagaacaat aggcctagcg gcatccccga gcggtttagc 180
ggctctaata gcggcaacac cgctaccctg actattagta gggctcaggc cggcgacgag 240
gccgactact actgtcagtc ctacgacggc cagaagtcac tggtctttgg cggcggaact 300
aagctgaccg tgctgggaca gcctaaggct gcccccagcg tgaccctgtt cccccccagc 360
agcgaggagc tgcaggccaa caaggccacc ctggtgtgcc tgatcagcga cttctaccca 420
ggcgccgtga ccgtggcctg gaaggccgac agcagccccg tgaaggccgg cgtggagacc 480
accaccccca gcaagcagag caacaacaag tacgccgcca gcagctacct gagcctgacc 540
cccgagcagt ggaagagcca caggtcctac agctgccagg tgacccacga gggcagcacc 600
gtggaaaaga ccgtggcccc aaccgagtgc agc 633
<210> 207
<211> 707
<212> PRT
<213> Homo Sapien
<400> 207
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Val Ile Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Ser Gly Val Asn Thr His Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Gly Ala Thr Ala Asn Asn Ile Arg Tyr Lys Phe Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
115 120 125
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
130 135 140
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
145 150 155 160
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
165 170 175
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
180 185 190
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
195 200 205
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro
210 215 220
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
225 230 235 240
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
290 295 300
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
305 310 315 320
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
325 330 335
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
340 345 350
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
355 360 365
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
370 375 380
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
385 390 395 400
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
405 410 415
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
420 425 430
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
435 440 445
Ser Leu Ser Pro Gly Lys Gly Gly Ser Gly Gly Ser Asp Ile Val Leu
450 455 460
Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr
465 470 475 480
Leu Ser Cys Arg Ala Ser Gln Phe Ile Gly Ser Arg Tyr Leu Ala Trp
485 490 495
Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Gly Ala
500 505 510
Ser Asn Arg Ala Thr Gly Val Pro Ala Arg Phe Ser Gly Ser Gly Ser
515 520 525
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu Asp Phe
530 535 540
Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Tyr Pro Gln Thr Phe Gly
545 550 555 560
Gln Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly
565 570 575
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu
580 585 590
Lys Glu Ser Gly Pro Ala Leu Val Lys Pro Thr Gln Thr Leu Thr Leu
595 600 605
Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Asn Arg Gly Gly Gly Val
610 615 620
Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu Trp Leu Ala Trp
625 630 635 640
Ile Asp Trp Asp Asp Asp Lys Ser Tyr Ser Thr Ser Leu Lys Thr Arg
645 650 655
Leu Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Val Val Leu Thr Met
660 665 670
Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr Cys Ala Arg Met
675 680 685
His Leu Pro Leu Val Phe Asp Ser Trp Gly Gln Gly Thr Leu Val Thr
690 695 700
Val Ser Ser
705
<210> 208
<211> 2121
<212> DNA
<213> Homo Sapien
<400> 208
gaggtgcagc tgctggaatc aggcggagga ctggtgcagc ctggcggatc actgagactg 60
agctgtgccg ctagtggctt cacctttagc gactatgtga ttaactgggt ccgacaggcc 120
cctggcaagg gactggaatg ggtgtcaggc attagttgga gcggcgtgaa cactcactac 180
gccgatagcg tgaagggccg gttcactatt agccgggata actctaagaa caccctgtac 240
ctgcagatga atagcctgag agccgaggac accgccgtct actactgcgc tagactgggc 300
gctaccgcta acaacatccg ctataagttc atggacgtgt ggggccaggg caccctggtc 360
acagtgtctt cagctagcac taagggcccc tcagtgttcc ccctggcccc tagctctaag 420
tctactagcg gtggcaccgc cgctctgggc tgcctggtca aggactactt ccccgagccc 480
gtgaccgtgt cttggaatag cggcgctctg actagcggag tgcacacctt ccccgccgtg 540
ctgcagtcta gcggcctgta tagcctgtct agcgtcgtga ccgtgcctag ctctagcctg 600
ggcactcaga cctatatctg taacgtgaac cacaagccta gtaacactaa ggtggacaag 660
cgggtggaac ctaagtcttg cgataagact cacacctgtc ccccctgccc tgccccagaa 720
gctgctggcg gacctagcgt gttcctgttc ccacctaagc ctaaagacac cctgatgatt 780
agtaggaccc ccgaagtgac ctgcgtggtg gtggacgtca gccacgagga ccctgaagtg 840
aagttcaatt ggtatgtgga cggcgtggaa gtgcacaacg ctaagactaa gcctagagag 900
gaacagtata actccaccta tagggtggtg tcagtgctga ccgtgctgca ccaggactgg 960
ctgaacggca aagagtataa gtgtaaagtc tctaacaagg ccctgcctgc ccctatcgaa 1020
aagactatct ctaaggctaa gggccagcct agagaacccc aggtctacac cctgccccct 1080
agtagagaag agatgactaa gaatcaggtg tccctgacct gtctggtcaa gggcttctac 1140
cctagcgata tcgccgtgga gtgggagtct aacggccagc ccgagaacaa ctataagact 1200
accccccctg tgctggatag cgacggctct ttcttcctgt actctaaact gaccgtggac 1260
aagtctaggt ggcagcaggg caacgtgttc agctgtagcg tgatgcacga ggccctgcac 1320
aatcactaca ctcagaagtc actgagcctg agtcccggca agggcggctc aggcggtagc 1380
gatatcgtgc tgactcagtc acccgctacc ctgagtctga gccctggcga gcgggctaca 1440
ctgagctgta gagctagtca gtttatcggc tcacgctacc tggcctggta tcagcagaag 1500
cccggccagg cccctagact gctgatctac ggcgctagta atagagctac cggcgtgccc 1560
gctaggttta gcggctcagg atcaggcacc gactttaccc tgactattag tagcctggaa 1620
cccgaggact tcgctaccta ctactgtcag cagtactacg actaccctca gaccttcggc 1680
cagggaacta aggtcgagat taagggcggt ggcggtagcg gcggaggcgg atcaggtggt 1740
ggtggtagtg gcggcggagg tagtcaggtc cagctgaaag agtcaggccc tgccctggtc 1800
aagcctactc agaccctgac cctgacctgc acttttagcg gctttagcct gagtaataga 1860
ggcggcggag tgggctggat tagacagcct ccaggcaaag ccctggagtg gctggcctgg 1920
atcgactggg acgacgataa gtcctactcc actagcctga aaactaggct gacaatcagc 1980
aaggacacta gtaaaaacca ggtggtgctg actatgacta atatggaccc cgtggacacc 2040
gctacctatt attgcgctag aatgcacctc ccactggtgt tcgatagctg gggtcaggga 2100
actctggtca cagtcagtag c 2121
Claims (108)
- 차단되지 않은 저밀도 지단백질-관련 단백질 6 (LRP6) 결합 단백질의 존재 하에 하나 이상의 LRP6 수용체를 한데 모음으로써 Wnt 신호를 강화하는, LRP6에 대한 단리된 이가 항체 또는 그의 이가 단편.
- 제1항에 있어서, 프로펠러 1 및 프로펠러 3으로 이루어진 군 중에서 선택된 프로펠러 영역에 대한 결합에 의해 하나 이상의 LRP6 수용체를 한데 모으는 단리된 이가 항체 또는 그의 이가 단편.
- 제2항에 있어서, LRP6 결합 단백질이 Wnt1, Wnt3, 및 Wnt3a로 이루어진 군 중에서 선택된 Wnt 결합 단백질인 단리된 이가 항체 또는 그의 이가 단편.
- 차단되지 않은 저밀도 지단백질-관련 단백질 6 (LRP6) 결합 단백질의 존재 하에 하나 이상의 LRP6 수용체를 한데 모음으로써 Wnt 신호의 강화를 방지하는, LRP6에 대한 단리된 일가 항체 또는 그의 일가 단편.
- 해리 속도 상수 (KD)가 적어도 1 x 107 M-1, 108 M-1, 109 M-1, 1010 M-1, 1011 M-1, 1012 M-1, 또는 1013 M-1인, 일가 또는 이가인, 저밀도 지단백질-관련 단백질 6 (LRP6) 단백질에 대한 단리된 항체 또는 그의 단편.
- 제5항에 있어서, 용액 평형 적정 검정에서 인간 LRP6에 대한 시험관내 결합에 의해 측정시에 정형적 Wnt 경로를 0.001 nM-1 μM의 KD로 억제하는, 일가 또는 이가인, 단리된 항체 또는 그의 단편.
- Wnt 리간드 유도 인산화 검정에 의해 평가시에 저밀도 지단백질-관련 단백질 6 (LRP6)의 인산화를 억제하는, 일가 또는 이가인, LRP6 단백질에 대한 단리된 항체 또는 그의 단편.
- 용액 평형 적정 (SET) 검정에서 인간 저밀도 지단백질-관련 단백질 6 (LRP6)에 대한 시험관내 결합에 의해 측정시에 정형적 Wnt 경로를 300 μM 이하의 EC50으로 억제하는, 일가 또는 이가인, LRP6 단백질에 대한 단리된 항체 또는 그의 단편.
- 표 1에 기재된 항체와 교차 경쟁하는, 일가 또는 이가인, LRP6 단백질에 대한 항체 또는 그의 단편.
- 표 1에 기재된 항체와 동일한 에피토프에 결합하는, 일가 또는 이가인 항체 또는 그의 단편.
- 제1항, 제4항, 제9항, 및 제10항 중 어느 한 항에 있어서, 모노클로날 항체인 항체.
- 제11항에 있어서, 인간 또는 인간화 항체인 항체.
- 제11항에 있어서, 키메라 항체인 항체.
- 제11항에 있어서, 인간 중쇄 불변 영역 및 인간 경쇄 불변 영역을 포함하는 항체.
- 제11항에 있어서, 단일 쇄 항체인 항체.
- 제11항에 있어서, Fab 단편인 항체.
- 제11항에 있어서, scFv인 항체.
- 제11항에 있어서, 인간 LRP6 및 시노몰구스 LRP6 둘 모두에 결합하는 항체.
- 제11항에 있어서, IgG 이소형인 항체.
- 제11항에 있어서, 각각의 인간 VH 또는 VL 배선 서열로부터의 항체 프레임워크로 치환된 아미노산을 갖는 프레임워크를 포함하는 항체.
- 표 1 내의 임의의 항체의 1, 2, 3, 4, 5, 또는 6개의 CDR을 포함하는, 일가 또는 이가인, LRP6에 대한 단리된 항체 또는 그의 단편.
- 서열 3, 서열 25, 서열 및 서열 43으로 이루어진 군 중에서 선택된 중쇄 CDR3을 포함하는, 일가 또는 이가인, LRP6에 대한 단리된 항체 또는 그의 단편.
- 서열 14, 서열 34, 서열 36, 서열 44, 서열 60, 및 서열 62로 이루어진 군 중에서 선택된 VH 및 서열 13, 서열 33, 서열 35, 서열 43, 서열 59, 및 서열 61로 이루어진 군 중에서 선택된 VL을 포함하는, 일가 또는 이가인, LRP6에 대한 단리된 항체 또는 그의 단편.
- 서열 14를 포함하는 VH 및 서열 13을 포함하는 VL을 포함하는, 일가 또는 이가인, LRP6에 대한 단리된 항체 또는 그의 단편.
- 서열 34를 포함하는 VH 및 서열 33을 포함하는 VL을 포함하는, 일가 또는 이가인, LRP6에 대한 단리된 항체 또는 그의 단편.
- 서열 36을 포함하는 VH 및 서열 35를 포함하는 VL을 포함하는, 일가 또는 이가인, LRP6에 대한 단리된 항체 또는 그의 단편.
- 서열 44를 포함하는 VH 및 서열 43을 포함하는 VL을 포함하는, 일가 또는 이가인, LRP6에 대한 단리된 항체 또는 그의 단편.
- 서열 36을 포함하는 VH 및 서열 35를 포함하는 VL을 포함하는, 일가 또는 이가인, LRP6에 대한 단리된 항체 또는 그의 단편.
- 서열 60을 포함하는 VH 및 서열 59를 포함하는 VL을 포함하는, 일가 또는 이가인, LRP6에 대한 단리된 항체 또는 그의 단편.
- 서열 61을 포함하는 VH 및 서열 62를 포함하는 VL을 포함하는, 일가 또는 이가인, LRP6에 대한 단리된 항체 또는 그의 단편.
- 서열 1, 2, 3, 21, 22, 23, 47, 48, 및 49와 동일한 적어도 하나의 중쇄 CDR 서열을 포함하고, 인간 LRP6 단백질에 결합하는, 단리된 항체 또는 그의 단편.
- 서열 4, 5, 6, 24, 25, 26, 50, 51 및 52와 동일한 적어도 하나의 경쇄 CDR 서열을 포함하고, 인간 LRP6 단백질에 결합하는, 일가 또는 이가인 단리된 모노클로날 항체 또는 그의 단편.
- 서열 1, 2, 3, 21, 22, 23, 47, 48, 및 49에 대해 적어도 95%의 서열 동일성을 갖는 적어도 하나의 중쇄 CDR 서열을 포함하고, 인간 LRP6 단백질에 결합하는, 일가 또는 이가인 단리된 항체 또는 그의 단편.
- 서열 4, 5, 6, 24, 25, 26, 50, 51 및 52에 대해 적어도 95%의 서열 동일성을 갖는 적어도 하나의 경쇄 CDR 서열을 포함하고, 인간 LRP6 단백질에 결합하는, 일가 또는 이가인 단리된 항체 또는 그의 단편.
- 서열 1의 중쇄 가변 영역 CDR1; 서열 2의 중쇄 가변 영역 CDR2; 서열 3의 중쇄 가변 영역 CDR3; 서열 4의 경쇄 가변 영역 CDR1; 서열 5의 경쇄 가변 영역 CDR2; 및 서열 6의 경쇄 가변 영역 CDR3을 포함하는, 일가 또는 이가인 단리된 항체 또는 그의 단편.
- 서열 21의 중쇄 가변 영역 CDR1; 서열 22의 중쇄 가변 영역 CDR2; 서열 23의 중쇄 가변 영역 CDR3; 서열 24의 경쇄 가변 영역 CDR1; 서열 25의 경쇄 가변 영역 CDR2; 및 서열 26의 경쇄 가변 영역 CDR3을 포함하는, 일가 또는 이가인 단리된 항체 또는 그의 단편.
- 서열 47의 중쇄 가변 영역 CDR1; 서열 48의 중쇄 가변 영역 CDR2; 서열 49의 중쇄 가변 영역 CDR3; 서열 50의 경쇄 가변 영역 CDR1; 서열 51의 경쇄 가변 영역 CDR2; 및 서열 52의 경쇄 가변 영역 CDR3을 포함하는, 일가 또는 이가인 단리된 항체 또는 그의 단편.
- Fab, F(ab2)', F(ab)2', scFv, VHH, VH, VL, dAb로 이루어진 군 중에서 선택되고, 일가 또는 이가인, LRP6에 결합하는 제1항 내지 제37항 중 어느 한 항에 따른 항체의 단편.
- 제1항 내지 제38항 중 어느 한 항에 따른 항체 또는 그의 단편 및 제약상 허용되는 담체를 포함하고, 여기서 항체 또는 그의 단편이 일가 또는 이가인, 제약 조성물.
- 서열 14, 34, 36, 44, 60, 및 62에 대해 중쇄 가변 영역 동일성을 포함하는 폴리펩티드를 코딩하는 뉴클레오티드 서열을 포함하는 핵산.
- 서열 13, 33, 35, 43, 59 및 61에 대해 경쇄 가변 영역 동일성을 포함하는 폴리펩티드를 코딩하는 뉴클레오티드 서열을 포함하는 핵산.
- 서열 14, 34, 36, 44, 60, 및 62에 대해 적어도 98%의 서열 동일성을 갖는 중쇄 가변 영역을 포함하는 폴리펩티드를 코딩하는 뉴클레오티드 서열을 포함하는 핵산.
- 서열 13, 33, 35, 43, 59 및 61에 대해 적어도 98%의 서열 동일성을 갖는 경쇄 가변 영역을 포함하는 폴리펩티드를 코딩하는 뉴클레오티드 서열을 포함하는 핵산.
- 제40항 내지 제43항 중 어느 한 항에 따른 핵산을 포함하는 벡터.
- 서열 69, 서열 93, 및 서열 115로 이루어진 군 중에서 선택된 CDR3을 포함하고, 일가 또는 이가인, LRP6에 대한 단리된 항체 또는 그의 단편.
- 서열 69, 서열 93, 및 서열 115로 이루어진 군 중에서 선택된 중쇄 CDR1; 서열 70, 서열 94, 및 서열 116으로 이루어진 군 중에서 선택된 CDR2; 및 서열 71, 서열 95, 및 서열 117로 이루어진 군 중에서 선택된 CDR3을 포함하고, 일가 또는 이가인, LRP6에 대한 단리된 항체 또는 그의 단편.
- 서열 72, 서열 96, 및 서열 118로 이루어진 군 중에서 선택된 경쇄 CDR1; 서열 73, 서열 97, 및 서열 119로 이루어진 군 중에서 선택된 CDR2; 및 서열 74, 서열 98, 및 서열 120으로 이루어진 군 중에서 선택된 CDR3을 포함하고, 일가 또는 이가인, LRP6에 대한 단리된 항체 또는 그의 단편.
- 서열 82, 서열 89, 서열 106, 서열 108, 서열 128, 서열 130, 및 서열 138로 이루어진 군 중에서 선택된 VH; 및 서열 81, 서열 90, 서열 105, 서열 107, 서열 127, 서열 129, 및 서열 137로 이루어진 군 중에서 선택된 VL을 포함하고, 일가 또는 이가인, LRP6에 대한 단리된 항체 또는 그의 단편.
- 서열 82를 포함하는 VH 및 서열 81을 포함하는 VL을 포함하고, 일가 또는 이가인, LRP6에 대한 단리된 항체 또는 그의 단편.
- 서열 90을 포함하는 VH 및 서열 89를 포함하는 VL을 포함하고, 일가 또는 이가인, LRP6에 대한 단리된 항체 또는 그의 단편.
- 서열 106을 포함하는 VH 및 서열 105를 포함하는 VL을 포함하고, 일가 또는 이가인, LRP6에 대한 단리된 항체 또는 그의 단편.
- 서열 108을 포함하는 VH 및 서열 107을 포함하는 VL을 포함하고, 일가 또는 이가인, LRP6에 대한 단리된 항체 또는 그의 단편.
- 서열 128을 포함하는 VH 및 서열 127을 포함하는 VL을 포함하고, 일가 또는 이가인, LRP6에 대한 단리된 항체 또는 그의 단편.
- 서열 128을 포함하는 VH 및 서열 127을 포함하는 VL을 포함하고, 일가 또는 이가인, LRP6에 대한 단리된 항체 또는 그의 단편.
- 서열 130을 포함하는 VH 및 서열 129를 포함하는 VL을 포함하고, 일가 또는 이가인, LRP6에 대한 단리된 항체 또는 그의 단편.
- 서열 138을 포함하는 VH 및 서열 137을 포함하는 VL을 포함하고, 일가 또는 이가인, LRP6에 대한 단리된 항체 또는 그의 단편.
- 서열 69, 70, 71, 93, 94, 95, 115, 116, 및 117과 동일한 적어도 하나의 중쇄 CDR 서열을 포함하고, 인간 LRP6 단백질에 결합하는 일가 또는 이가인 단리된 항체 또는 그의 단편.
- 서열 72, 73, 74, 96, 97, 98, 118, 119, 및 120과 동일한 적어도 하나의 경쇄 CDR 서열을 포함하고, 인간 LRP6 단백질에 결합하는 일가 또는 이가인 단리된 항체 또는 그의 단편.
- 서열 70, 71, 93, 94, 95, 115, 116, 및 117에 대해 적어도 95%의 서열 동일성을 갖는 적어도 하나의 중쇄 CDR 서열을 포함하고, 인간 LRP6 단백질에 결합하는 일가 또는 이가인 단리된 항체 또는 그의 단편.
- 서열 72, 73, 74, 96, 97, 98, 118, 119, 및 120에 대해 적어도 95%의 서열 동일성을 갖는 적어도 하나의 경쇄 CDR 서열을 포함하고, 인간 LRP6 단백질에 결합하는 일가 또는 이가인 단리된 항체 또는 그의 단편.
- 서열 69의 중쇄 가변 영역 CDR1; 서열 70의 중쇄 가변 영역 CDR2; 서열 71의 중쇄 가변 영역 CDR3; 서열 72의 경쇄 가변 영역 CDR1; 서열 73의 경쇄 가변 영역 CDR2; 및 서열 74의 경쇄 가변 영역 CDR3을 포함하는 일가 또는 이가인 단리된 항체 또는 그의 단편.
- 서열 93의 중쇄 가변 영역 CDR1; 서열 94의 중쇄 가변 영역 CDR2; 서열 95의 중쇄 가변 영역 CDR3; 서열 96의 경쇄 가변 영역 CDR1; 서열 97의 경쇄 가변 영역 CDR2; 및 서열 98의 경쇄 가변 영역 CDR3을 포함하는 일가 또는 이가인 단리된 항체 또는 그의 단편.
- 서열 115의 중쇄 가변 영역 CDR1; 서열 116의 중쇄 가변 영역 CDR2; 서열 117의 중쇄 가변 영역 CDR3; 서열 118의 경쇄 가변 영역 CDR1; 서열 119의 경쇄 가변 영역 CDR2; 및 서열 120의 경쇄 가변 영역 CDR3을 포함하는 일가 또는 이가인 단리된 항체 또는 그의 단편.
- 제35항 내지 제37항 중 어느 한 항에 따른 항체로 이루어진 군 중에서 선택된 모노클로날 항체 또는 그의 단편과 제65항 내지 제63항 중 어느 한 항에 따른 항체와의 조합물이며, 여기서 항체 또는 그의 단편이 일가인, 조합물.
- 제35항 내지 제37항 중 어느 한 항에 따른 항체로 이루어진 군 중에서 선택된 모노클로날 항체 또는 그의 단편과 제65항 내지 제63항 중 어느 한 항에 따른 항체와의 조합물이며, 여기서 항체 또는 그의 단편이 이가인, 조합물.
- Fab, F(ab2)', F(ab)2', scFv, VHH, VH, VL, dAb로 이루어진 군 중에서 선택되고, 일가 또는 이가인, LRP6에 결합하는 제1항 내지 제65항 중 어느 한 항에 따른 항체의 단편.
- 제45항 내지 제63항 중 어느 한 항에 따른 항체 및 제약상 허용되는 담체를 포함하고, 여기서 항체가 일가 또는 이가인, 제약 조성물.
- 서열 82, 89, 106, 108, 128, 130, 및 138로 이루어진 군 중에서 선택된 중쇄 가변 영역을 포함하는 폴리펩티드를 코딩하는 뉴클레오티드 서열을 포함하는 핵산.
- 서열 81, 90, 105, 107, 127, 129, 및 137로 이루어진 군 중에서 선택된 경쇄 가변 영역을 포함하는 폴리펩티드를 코딩하는 뉴클레오티드 서열을 포함하는 핵산.
- 서열 82, 89, 106, 108, 128, 130, 및 138에 대해 적어도 98%의 서열 동일성을 갖는 중쇄 가변 영역을 포함하는 폴리펩티드를 코딩하는 뉴클레오티드 서열을 포함하는 핵산.
- 서열 81, 90, 105, 107, 127, 129, 및 137에 대해 적어도 98%의 서열 동일성을 갖는 경쇄 가변 영역을 포함하는 폴리펩티드를 코딩하는 뉴클레오티드 서열을 포함하는 핵산.
- 제68항 내지 제71항 중 어느 한 항에 따른 핵산을 포함하는 벡터.
- LRP6 발현 암이 있는 대상체를 선택하고, 상기 대상체에게 제1항 내지 제38항 및 제45항 내지 제65항 중 어느 한 항에 따른 항체 또는 그의 단편을 포함하는 유효량의 조성물을 투여하는 것을 포함하고, 여기서 항체 또는 그의 단편이 일가 또는 이가인, 암의 치료 방법.
- 제73항에 있어서, 대상체가 인간인 방법.
- LRP6 발현 암이 있는 대상체를 선택하고, 상기 대상체에게 제1항 내지 제39항 및 제45항 내지 제66항 중 어느 한 항에 따른 항체 또는 그의 단편을 포함하는 유효량의 조성물을 투여하는 것을 포함하고, 여기서 항체 또는 그의 단편이 일가 또는 이가이고, 상기 암이 유방암, 폐암, 다발성 골수종, 난소암, 간암, 방광암, 위암, 전립선암, 급성 골수성 백혈병, 만성 골수성 백혈병, 골육종, 편평세포 암종, 및 흑색종으로 이루어진 군 중에서 선택된 것인, 정형적 Wnt 신호 전달 경로에 의해 매개되는 암의 치료 방법.
- 제75항에 있어서, 상기 암이 유방암인 방법.
- LRP6 발현 암이 있는 대상체를 선택하고, 상기 대상체에게 LRP6의 프로펠러 1 영역에 결합하는 항체 또는 그의 단편을 LRP6의 프로펠러 3 영역에 결합하는 항체 또는 그의 단편과 조합하여 포함하는 유효량의 조성물을 투여하는 것을 포함하고, 여기서 항체 또는 그의 단편이 일가 또는 이가인 암의 치료 방법.
- LRP6 발현 암이 있는 대상체를 선택하고, 상기 대상체에게 LRP6에 결합하여 LRP6에 의한 Wnt1 신호 전달을 억제하는 항체 또는 그의 단편을 LRP6에 결합하여 LRP6에 의한 Wnt3 신호 전달을 억제하는 항체 또는 그의 단편과 조합하여 포함하는 유효량의 조성물을 투여하는 것을 포함하고, 여기서 항체 또는 그의 단편이 일가 또는 이가인 암의 치료 방법.
- 유방암, 폐암, 다발성 골수종, 난소암, 간암, 방광암, 위암, 전립선암, 급성 골수성 백혈병, 만성 골수성 백혈병, 골육종, 편평세포 암종, 및 흑색종으로 이루어진 군 중에서 선택된, 정형적 Wnt 신호 전달 경로에 의해 매개되는 암의 치료를 위한 의약의 제조에 있어서 일가 또는 이가인 제1항 내지 제78항 중 어느 한 항에 따른 항체 또는 그의 단편의 용도.
- 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한 것이며, 일가 또는 이가인, 서열 14의 VH 및 서열 13의 VL을 갖는 항체.
- 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한 것이며, 일가 또는 이가인, 서열 34의 VH 및 서열 36의 VL을 갖는 항체.
- 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한 것이며, 일가 또는 이가인, 서열 36의 VH 및 서열 35의 VL을 갖는 항체.
- 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한 것이며, 일가 또는 이가인, 서열 44의 VH 및 서열 43의 VL을 갖는 항체.
- 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한 것이며, 일가 또는 이가인, 서열 60의 VH 및 서열 59의 VL을 갖는 항체.
- 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한 것이며, 일가 또는 이가인, 서열 62의 VH 및 서열 61의 VL을 갖는 항체.
- 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한 것이며, 일가 또는 이가인, 서열 82의 VH 및 서열 81의 VL을 갖는 항체.
- 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한 것이며, 일가 또는 이가인, 서열 90의 VH 및 서열 89의 VL을 갖는 항체.
- 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한 것이며, 일가 또는 이가인, 서열 106의 VH 및 서열 105의 VL을 갖는 항체.
- 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한 것이며, 일가 또는 이가인, 서열 108의 VH 및 서열 107의 VL을 갖는 항체.
- 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한 것이며, 일가 또는 이가인, 서열 128의 VH 및 서열 127의 VL을 갖는 항체.
- 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한 것이며, 일가 또는 이가인, 서열 130의 VH 및 서열 129의 VL을 갖는 항체.
- 정형적 Wnt 신호 전달 경로에 의해 매개되는 암을 치료하는데 사용하기 위한 것이며, 일가 또는 이가인, 서열 138의 VH 및 서열 137의 VL을 갖는 항체.
- 약물로서 사용하기 위한 것이며, 일가 또는 이가인, 서열 14의 VH 및 서열 13의 VL을 갖는 항체.
- 약물로서 사용하기 위한 것이며, 일가 또는 이가인, 서열 34의 VH 및 서열 33의 VL을 갖는 항체.
- 약물로서 사용하기 위한 것이며, 일가 또는 이가인, 서열 36의 VH 및 서열 35의 VL을 갖는 항체.
- 약물로서 사용하기 위한 것이며, 일가 또는 이가인, 서열 44의 VH 및 서열 43의 VL을 갖는 항체.
- 약물로서 사용하기 위한 것이며, 일가 또는 이가인, 서열 60의 VH 및 서열 59의 VL을 갖는 항체.
- 약물로서 사용하기 위한 것이며, 일가 또는 이가인, 서열 62의 VH 및 서열 61의 VL을 갖는 항체.
- 약물로서 사용하기 위한 것이며, 일가 또는 이가인, 서열 82의 VH 및 서열 81의 VL을 갖는 항체.
- 약물로서 사용하기 위한 것이며, 일가 또는 이가인, 서열 90의 VH 및 서열 89의 VL을 갖는 항체.
- 약물로서 사용하기 위한 것이며, 일가 또는 이가인, 서열 106의 VH 및 서열 105의 VL을 갖는 항체.
- 약물로서 사용하기 위한 것이며, 일가 또는 이가인, 서열 108의 VH 및 서열 107의 VL을 갖는 항체.
- 약물로서 사용하기 위한 것이며, 일가 또는 이가인, 서열 128의 VH 및 서열 127의 VL을 갖는 항체.
- 약물로서 사용하기 위한 것이며, 일가 또는 이가인, 서열 130의 VH 및 서열 129의 VL을 갖는 항체.
- 약물로서 사용하기 위한 것이며, 일가 또는 이가인, 서열 138의 VH 및 서열 137의 VL을 갖는 항체.
- 의약으로서 사용하기 위한, 제1항 내지 제38항 및 제45항 내지 제66항 중 어느 한 항에 따른 항체 또는 그의 단편.
- LRP6 발현 암의 치료를 위한 의약으로서 사용하기 위한, 제1항 내지 제38항 및 제45항 내지 제66항 중 어느 한 항에 따른 항체 또는 그의 단편.
- 유방암, 폐암, 다발성 골수종, 난소암, 간암, 방광암, 위암, 전립선암, 급성 골수성 백혈병, 만성 골수성 백혈병, 골육종, 편평세포 암종, 및 흑색종으로 이루어진 군 중에서 선택된 LRP6 발현 암의 치료를 위한 의약으로서 사용하기 위한, 제1항 내지 제38항 및 제45항 내지 제66항 중 어느 한 항에 따른 항체 또는 그의 단편.
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US33198510P | 2010-05-06 | 2010-05-06 | |
| US61/331,985 | 2010-05-06 | ||
| PCT/EP2011/057202 WO2011138392A1 (en) | 2010-05-06 | 2011-05-05 | Compositions and methods of use for therapeutic low density lipoprotein -related protein 6 (lrp6) antibodies |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20130066632A true KR20130066632A (ko) | 2013-06-20 |
Family
ID=44227756
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020127031870A KR20130066632A (ko) | 2010-05-06 | 2011-05-05 | 치료적 저밀도 지단백질-관련 단백질 6 (lrp6) 항체에 대한 조성물 및 사용 방법 |
Country Status (22)
| Country | Link |
|---|---|
| US (3) | US9428583B2 (ko) |
| EP (3) | EP3345926B1 (ko) |
| JP (5) | JP2013527762A (ko) |
| KR (1) | KR20130066632A (ko) |
| CN (1) | CN103038258B (ko) |
| AU (2) | AU2011249783B9 (ko) |
| BR (1) | BR112012028306A2 (ko) |
| CA (1) | CA2798432A1 (ko) |
| CL (2) | CL2012003094A1 (ko) |
| CO (1) | CO6630181A2 (ko) |
| EA (1) | EA201291180A1 (ko) |
| ES (2) | ES2659406T3 (ko) |
| GT (1) | GT201200297A (ko) |
| HK (1) | HK1258029A1 (ko) |
| IL (1) | IL222811A0 (ko) |
| MX (1) | MX2012012928A (ko) |
| NZ (1) | NZ603829A (ko) |
| PE (1) | PE20130207A1 (ko) |
| SG (1) | SG185416A1 (ko) |
| TN (1) | TN2012000511A1 (ko) |
| WO (1) | WO2011138392A1 (ko) |
| ZA (1) | ZA201208221B (ko) |
Families Citing this family (17)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| PL2567709T3 (pl) * | 2007-11-02 | 2018-06-29 | Novartis Ag | Cząsteczki i sposoby modulowania białka związanego z receptorem dla lipoproteiny o niskiej gęstości 6 (LRP6) |
| NZ602040A (en) | 2010-03-24 | 2014-12-24 | Genentech Inc | Anti-lrp6 antibodies |
| ES2659406T3 (es) | 2010-05-06 | 2018-03-15 | Novartis Ag | Composiciones y procedimientos de uso para anticuerpos terapéuticos contra la proteína 6 relacionada con las lipoproteínas de baja densidad (LRP6) |
| AU2011249782B2 (en) * | 2010-05-06 | 2014-10-02 | Novartis Ag | Compositions and methods of use for therapeutic low density lipoprotein - related protein 6 (LRP6) multivalent antibodies |
| EP2773669B1 (en) | 2011-11-04 | 2018-03-28 | Novartis AG | Low density lipoprotein-related protein 6 (lrp6) - half life extender constructs |
| US10562946B2 (en) | 2014-06-20 | 2020-02-18 | Genentech, Inc. | Chagasin-based scaffold compositions, methods, and uses |
| WO2016040895A1 (en) | 2014-09-12 | 2016-03-17 | xxTHE BOARD OF TRUSTEES OF THE LELAND STANFORD JUNIOR UNIVERSITY | Wnt signaling agonist molecules |
| ES2821099T3 (es) | 2015-12-04 | 2021-04-23 | Boehringer Ingelheim Int Gmbh & Co Kg | Polipéptidos biparatópicos antagonistas de la señalización WNT en células tumorales |
| CN107029211A (zh) * | 2017-05-05 | 2017-08-11 | 上海市东方医院(同济大学附属东方医院) | 一种用于治疗心衰保护心脏功能的药物 |
| EP3630816B1 (en) * | 2017-05-31 | 2024-03-20 | Boehringer Ingelheim International GmbH | Polypeptides antagonizing wnt signaling in tumor cells |
| US11773171B2 (en) | 2017-12-19 | 2023-10-03 | Surrozen Operating, Inc. | WNT surrogate molecules and uses thereof |
| US11746150B2 (en) | 2017-12-19 | 2023-09-05 | Surrozen Operating, Inc. | Anti-LRP5/6 antibodies and methods of use |
| CA3130663A1 (en) * | 2019-03-29 | 2020-10-08 | Boehringer Ingelheim International Gmbh | Anticancer combination therapy |
| WO2020200998A1 (en) * | 2019-03-29 | 2020-10-08 | Boehringer Ingelheim International Gmbh | Anticancer combination therapy |
| JP2022543669A (ja) * | 2019-08-08 | 2022-10-13 | リジェネロン・ファーマシューティカルズ・インコーポレイテッド | 新規抗原結合分子フォーマット |
| WO2021026666A1 (en) * | 2019-08-14 | 2021-02-18 | Modmab Therapeutics Inc. | Antibodies that bind to lrp6 proteins and methods of use |
| WO2023147293A2 (en) * | 2022-01-25 | 2023-08-03 | The Trustees Of The University Of Pennsylvania | Compositions and methods comprising anti-cd38 chimeric antigen receptors (cars) |
Family Cites Families (216)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US2207308A (en) | 1940-07-09 | Electrolytic manufacture | ||
| US4458066A (en) | 1980-02-29 | 1984-07-03 | University Patents, Inc. | Process for preparing polynucleotides |
| ATE37983T1 (de) | 1982-04-22 | 1988-11-15 | Ici Plc | Mittel mit verzoegerter freigabe. |
| US4522811A (en) | 1982-07-08 | 1985-06-11 | Syntex (U.S.A.) Inc. | Serial injection of muramyldipeptides and liposomes enhances the anti-infective activity of muramyldipeptides |
| CA1247080A (en) | 1983-03-08 | 1988-12-20 | Commonwealth Serum Laboratories Commission | Antigenically active amino acid sequences |
| US4816567A (en) | 1983-04-08 | 1989-03-28 | Genentech, Inc. | Recombinant immunoglobin preparations |
| DE3572982D1 (en) | 1984-03-06 | 1989-10-19 | Takeda Chemical Industries Ltd | Chemically modified lymphokine and production thereof |
| US5128326A (en) | 1984-12-06 | 1992-07-07 | Biomatrix, Inc. | Drug delivery systems based on hyaluronans derivatives thereof and their salts and methods of producing same |
| US5374548A (en) | 1986-05-02 | 1994-12-20 | Genentech, Inc. | Methods and compositions for the attachment of proteins to liposomes using a glycophospholipid anchor |
| MX9203291A (es) | 1985-06-26 | 1992-08-01 | Liposome Co Inc | Metodo para acoplamiento de liposomas. |
| US5225539A (en) | 1986-03-27 | 1993-07-06 | Medical Research Council | Recombinant altered antibodies and methods of making altered antibodies |
| US5260203A (en) | 1986-09-02 | 1993-11-09 | Enzon, Inc. | Single polypeptide chain binding molecules |
| US4881175A (en) | 1986-09-02 | 1989-11-14 | Genex Corporation | Computer based system and method for determining and displaying possible chemical structures for converting double- or multiple-chain polypeptides to single-chain polypeptides |
| WO1988007089A1 (en) | 1987-03-18 | 1988-09-22 | Medical Research Council | Altered antibodies |
| US5013653A (en) | 1987-03-20 | 1991-05-07 | Creative Biomolecules, Inc. | Product and process for introduction of a hinge region into a fusion protein to facilitate cleavage |
| EP0623679B1 (en) | 1987-05-21 | 2003-06-25 | Micromet AG | Targeted multifunctional proteins |
| US5132405A (en) | 1987-05-21 | 1992-07-21 | Creative Biomolecules, Inc. | Biosynthetic antibody binding sites |
| US5258498A (en) | 1987-05-21 | 1993-11-02 | Creative Biomolecules, Inc. | Polypeptide linkers for production of biosynthetic proteins |
| US5091513A (en) | 1987-05-21 | 1992-02-25 | Creative Biomolecules, Inc. | Biosynthetic antibody binding sites |
| US4880078A (en) | 1987-06-29 | 1989-11-14 | Honda Giken Kogyo Kabushiki Kaisha | Exhaust muffler |
| US5677425A (en) | 1987-09-04 | 1997-10-14 | Celltech Therapeutics Limited | Recombinant antibody |
| US5336603A (en) | 1987-10-02 | 1994-08-09 | Genentech, Inc. | CD4 adheson variants |
| DE3813023A1 (de) | 1988-04-19 | 1989-11-16 | Behringwerke Ag | Monoklonaler antikoerper gegen pseudomonas aeruginosa, seine herstellung und verwendung |
| US5476996A (en) | 1988-06-14 | 1995-12-19 | Lidak Pharmaceuticals | Human immune system in non-human animal |
| US5223409A (en) | 1988-09-02 | 1993-06-29 | Protein Engineering Corp. | Directed evolution of novel binding proteins |
| GB8823869D0 (en) | 1988-10-12 | 1988-11-16 | Medical Res Council | Production of antibodies |
| KR900005995A (ko) | 1988-10-31 | 1990-05-07 | 우메모또 요시마사 | 변형 인터류킨-2 및 그의 제조방법 |
| JP2919890B2 (ja) | 1988-11-11 | 1999-07-19 | メディカル リサーチ カウンスル | 単一ドメインリガンド、そのリガンドからなる受容体、その製造方法、ならびにそのリガンドおよび受容体の使用 |
| JP2989002B2 (ja) | 1988-12-22 | 1999-12-13 | キリン―アムジエン・インコーポレーテツド | 化学修飾顆粒球コロニー刺激因子 |
| US5530101A (en) | 1988-12-28 | 1996-06-25 | Protein Design Labs, Inc. | Humanized immunoglobulins |
| US5108921A (en) | 1989-04-03 | 1992-04-28 | Purdue Research Foundation | Method for enhanced transmembrane transport of exogenous molecules |
| ATE92107T1 (de) | 1989-04-29 | 1993-08-15 | Delta Biotechnology Ltd | N-terminale fragmente von menschliches serumalbumin enthaltenden fusionsproteinen. |
| US6291158B1 (en) | 1989-05-16 | 2001-09-18 | Scripps Research Institute | Method for tapping the immunological repertoire |
| US5112946A (en) | 1989-07-06 | 1992-05-12 | Repligen Corporation | Modified pf4 compositions and methods of use |
| FR2650598B1 (fr) | 1989-08-03 | 1994-06-03 | Rhone Poulenc Sante | Derives de l'albumine a fonction therapeutique |
| WO1991005548A1 (en) | 1989-10-10 | 1991-05-02 | Pitman-Moore, Inc. | Sustained release composition for macromolecular proteins |
| WO1991006570A1 (en) | 1989-10-25 | 1991-05-16 | The University Of Melbourne | HYBRID Fc RECEPTOR MOLECULES |
| JP2571874B2 (ja) | 1989-11-06 | 1997-01-16 | アルカーメス コントロールド セラピューティクス,インコーポレイテッド | タンパク質マイクロスフェア組成物 |
| DE69133566T2 (de) | 1990-01-12 | 2007-12-06 | Amgen Fremont Inc. | Bildung von xenogenen Antikörpern |
| US6150584A (en) | 1990-01-12 | 2000-11-21 | Abgenix, Inc. | Human antibodies derived from immunized xenomice |
| US6075181A (en) | 1990-01-12 | 2000-06-13 | Abgenix, Inc. | Human antibodies derived from immunized xenomice |
| US6673986B1 (en) | 1990-01-12 | 2004-01-06 | Abgenix, Inc. | Generation of xenogeneic antibodies |
| US5427908A (en) | 1990-05-01 | 1995-06-27 | Affymax Technologies N.V. | Recombinant library screening methods |
| US5349053A (en) | 1990-06-01 | 1994-09-20 | Protein Design Labs, Inc. | Chimeric ligand/immunoglobulin molecules and their uses |
| US6172197B1 (en) | 1991-07-10 | 2001-01-09 | Medical Research Council | Methods for producing members of specific binding pairs |
| GB9015198D0 (en) | 1990-07-10 | 1990-08-29 | Brien Caroline J O | Binding substance |
| US5625126A (en) | 1990-08-29 | 1997-04-29 | Genpharm International, Inc. | Transgenic non-human animals for producing heterologous antibodies |
| US5770429A (en) | 1990-08-29 | 1998-06-23 | Genpharm International, Inc. | Transgenic non-human animals capable of producing heterologous antibodies |
| US5545806A (en) | 1990-08-29 | 1996-08-13 | Genpharm International, Inc. | Ransgenic non-human animals for producing heterologous antibodies |
| US5877397A (en) | 1990-08-29 | 1999-03-02 | Genpharm International Inc. | Transgenic non-human animals capable of producing heterologous antibodies of various isotypes |
| US5814318A (en) | 1990-08-29 | 1998-09-29 | Genpharm International Inc. | Transgenic non-human animals for producing heterologous antibodies |
| US6300129B1 (en) | 1990-08-29 | 2001-10-09 | Genpharm International | Transgenic non-human animals for producing heterologous antibodies |
| US5661016A (en) | 1990-08-29 | 1997-08-26 | Genpharm International Inc. | Transgenic non-human animals capable of producing heterologous antibodies of various isotypes |
| US5633425A (en) | 1990-08-29 | 1997-05-27 | Genpharm International, Inc. | Transgenic non-human animals capable of producing heterologous antibodies |
| DK0814159T3 (da) | 1990-08-29 | 2005-10-24 | Genpharm Int | Transgene, ikke-humane dyr, der er i stand til at danne heterologe antistoffer |
| US6255458B1 (en) | 1990-08-29 | 2001-07-03 | Genpharm International | High affinity human antibodies and human antibodies against digoxin |
| US5874299A (en) | 1990-08-29 | 1999-02-23 | Genpharm International, Inc. | Transgenic non-human animals capable of producing heterologous antibodies |
| US5789650A (en) | 1990-08-29 | 1998-08-04 | Genpharm International, Inc. | Transgenic non-human animals for producing heterologous antibodies |
| US5242813A (en) | 1990-10-12 | 1993-09-07 | The United States Of America As Represented By The Department Of Health And Human Services | Mouse monoclonal antibodies specific for normal primate tissue, malignant human cultural cell lines human tumors |
| EP0517895B1 (en) | 1990-12-14 | 1996-11-20 | Cell Genesys, Inc. | Chimeric chains for receptor-associated signal transduction pathways |
| ES2181673T3 (es) | 1991-05-01 | 2003-03-01 | Jackson H M Found Military Med | Procedimiento de tratamiento de las enfermedades respiratorias infecciosas. |
| ATE463573T1 (de) | 1991-12-02 | 2010-04-15 | Medimmune Ltd | Herstellung von autoantikörpern auf phagenoberflächen ausgehend von antikörpersegmentbibliotheken |
| JP3949712B2 (ja) | 1991-12-02 | 2007-07-25 | メディカル リサーチ カウンシル | 抗体セグメントレパートリー由来でファージに表示される抗自己抗体の産生 |
| JPH07503132A (ja) | 1991-12-17 | 1995-04-06 | ジェンファーム インターナショナル,インコーポレイティド | 異種抗体を産生することができるトランスジェニック非ヒト動物 |
| US5622929A (en) | 1992-01-23 | 1997-04-22 | Bristol-Myers Squibb Company | Thioether conjugates |
| FR2686901A1 (fr) | 1992-01-31 | 1993-08-06 | Rhone Poulenc Rorer Sa | Nouveaux polypeptides antithrombotiques, leur preparation et compositions pharmaceutiques les contenant. |
| FR2686899B1 (fr) | 1992-01-31 | 1995-09-01 | Rhone Poulenc Rorer Sa | Nouveaux polypeptides biologiquement actifs, leur preparation et compositions pharmaceutiques les contenant. |
| WO1993016178A2 (en) | 1992-02-12 | 1993-08-19 | The United States Of America, As Represented By The Secretary, Department Of Health And Human Services | Sequences characteristic of human gene transcription product |
| US5714350A (en) | 1992-03-09 | 1998-02-03 | Protein Design Labs, Inc. | Increasing antibody affinity by altering glycosylation in the immunoglobulin variable region |
| US5912015A (en) | 1992-03-12 | 1999-06-15 | Alkermes Controlled Therapeutics, Inc. | Modulated release from biocompatible polymers |
| US5447851B1 (en) | 1992-04-02 | 1999-07-06 | Univ Texas System Board Of | Dna encoding a chimeric polypeptide comprising the extracellular domain of tnf receptor fused to igg vectors and host cells |
| CA2118508A1 (en) | 1992-04-24 | 1993-11-11 | Elizabeth S. Ward | Recombinant production of immunoglobulin-like domains in prokaryotic cells |
| PT1498427E (pt) | 1992-08-21 | 2010-03-22 | Univ Bruxelles | Imunoglobulinas desprovidas de cadeias leves |
| US6765087B1 (en) | 1992-08-21 | 2004-07-20 | Vrije Universiteit Brussel | Immunoglobulins devoid of light chains |
| US5934272A (en) | 1993-01-29 | 1999-08-10 | Aradigm Corporation | Device and method of creating aerosolized mist of respiratory drug |
| WO1994021293A1 (en) | 1993-03-19 | 1994-09-29 | Duke University | Method of treatment of tumors with an antibody binding to tenascin |
| EP0754225A4 (en) | 1993-04-26 | 2001-01-31 | Genpharm Int | HETEROLOGIC ANTIBODY-PRODUCING TRANSGENIC NON-HUMAN ANIMALS |
| DK0698097T3 (da) | 1993-04-29 | 2001-10-08 | Unilever Nv | Produktion af antistoffer eller (funktionaliserede) fragmenter deraf afledt af Camelidae-immunoglobuliner med tung kæde |
| CA2163345A1 (en) | 1993-06-16 | 1994-12-22 | Susan Adrienne Morgan | Antibodies |
| SE9400088D0 (sv) | 1994-01-14 | 1994-01-14 | Kabi Pharmacia Ab | Bacterial receptor structures |
| US5605793A (en) | 1994-02-17 | 1997-02-25 | Affymax Technologies N.V. | Methods for in vitro recombination |
| US5834252A (en) | 1995-04-18 | 1998-11-10 | Glaxo Group Limited | End-complementary polymerase reaction |
| US5837458A (en) | 1994-02-17 | 1998-11-17 | Maxygen, Inc. | Methods and compositions for cellular and metabolic engineering |
| AU3382595A (en) | 1994-07-29 | 1996-03-04 | Smithkline Beecham Corporation | Novel compounds |
| US6132764A (en) | 1994-08-05 | 2000-10-17 | Targesome, Inc. | Targeted polymerized liposome diagnostic and treatment agents |
| JPH10511957A (ja) | 1995-01-05 | 1998-11-17 | ザ ボード オブ リージェンツ オブ ザ ユニヴァーシティ オブ ミシガン | 表面改質ナノ微粒子並びにその製造及び使用方法 |
| US5731168A (en) | 1995-03-01 | 1998-03-24 | Genentech, Inc. | Method for making heteromultimeric polypeptides |
| US5641870A (en) | 1995-04-20 | 1997-06-24 | Genentech, Inc. | Low pH hydrophobic interaction chromatography for antibody purification |
| US6019968A (en) | 1995-04-14 | 2000-02-01 | Inhale Therapeutic Systems, Inc. | Dispersible antibody compositions and methods for their preparation and use |
| US6121022A (en) | 1995-04-14 | 2000-09-19 | Genentech, Inc. | Altered polypeptides with increased half-life |
| US5869046A (en) | 1995-04-14 | 1999-02-09 | Genentech, Inc. | Altered polypeptides with increased half-life |
| AU710347B2 (en) | 1995-08-31 | 1999-09-16 | Alkermes Controlled Therapeutics, Inc. | Composition for sustained release of an agent |
| DK0885002T3 (da) | 1996-03-04 | 2011-08-22 | Penn State Res Found | Materialer og fremgangsmåder til forøgelse af cellulær internalisering |
| CA2249195A1 (en) | 1996-03-18 | 1997-09-25 | Board Of Regents, The University Of Texas System | Immunoglobin-like domains with increased half lives |
| US5874064A (en) | 1996-05-24 | 1999-02-23 | Massachusetts Institute Of Technology | Aerodynamically light particles for pulmonary drug delivery |
| US5855913A (en) | 1997-01-16 | 1999-01-05 | Massachusetts Instite Of Technology | Particles incorporating surfactants for pulmonary drug delivery |
| US5985309A (en) | 1996-05-24 | 1999-11-16 | Massachusetts Institute Of Technology | Preparation of particles for inhalation |
| US6056973A (en) | 1996-10-11 | 2000-05-02 | Sequus Pharmaceuticals, Inc. | Therapeutic liposome composition and method of preparation |
| WO1998023289A1 (en) | 1996-11-27 | 1998-06-04 | The General Hospital Corporation | MODULATION OF IgG BINDING TO FcRn |
| ES2236832T3 (es) | 1997-01-16 | 2005-07-16 | Massachusetts Institute Of Technology | Preparacion de particulas para inhalacion. |
| US6277375B1 (en) | 1997-03-03 | 2001-08-21 | Board Of Regents, The University Of Texas System | Immunoglobulin-like domains with increased half-lives |
| AU733722B2 (en) | 1997-04-15 | 2001-05-24 | Merck Sharp & Dohme Corp. | Novel LDL-receptor |
| EP0983303B1 (en) | 1997-05-21 | 2006-03-08 | Biovation Limited | Method for the production of non-immunogenic proteins |
| JP3614866B2 (ja) | 1997-06-12 | 2005-01-26 | リサーチ コーポレイション テクノロジーズ,インコーポレイティド | 人工抗体ポリペプチド |
| US5989463A (en) | 1997-09-24 | 1999-11-23 | Alkermes Controlled Therapeutics, Inc. | Methods for fabricating polymer-based controlled release devices |
| DE19742706B4 (de) | 1997-09-26 | 2013-07-25 | Pieris Proteolab Ag | Lipocalinmuteine |
| GB9722131D0 (en) | 1997-10-20 | 1997-12-17 | Medical Res Council | Method |
| SE512663C2 (sv) | 1997-10-23 | 2000-04-17 | Biogram Ab | Inkapslingsförfarande för aktiv substans i en bionedbrytbar polymer |
| US6780609B1 (en) | 1998-10-23 | 2004-08-24 | Genome Therapeutics Corporation | High bone mass gene of 1.1q13.3 |
| US6770461B1 (en) | 1998-10-23 | 2004-08-03 | Genome Therapeutics Corporation | High bone mass gene of 11q13.3 |
| US6194551B1 (en) | 1998-04-02 | 2001-02-27 | Genentech, Inc. | Polypeptide variants |
| EP2261229A3 (en) | 1998-04-20 | 2011-03-23 | GlycArt Biotechnology AG | Glycosylation engineering of antibodies for improving antibody-dependent cellular cytotoxicity |
| AU747231B2 (en) | 1998-06-24 | 2002-05-09 | Alkermes, Inc. | Large porous particles emitted from an inhaler |
| CA2339891A1 (en) | 1998-08-07 | 2000-02-17 | Onyx Pharmaceuticals, Inc. | Chp polypeptide, a ligand of pak65 |
| GB9824632D0 (en) | 1998-11-10 | 1999-01-06 | Celltech Therapeutics Ltd | Biological compounds |
| US6818418B1 (en) | 1998-12-10 | 2004-11-16 | Compound Therapeutics, Inc. | Protein scaffolds for antibody mimics and other binding proteins |
| NZ539776A (en) | 1999-01-15 | 2006-12-22 | Genentech Inc | Polypeptide variants with altered effector function |
| EP1161442A2 (en) | 1999-01-20 | 2001-12-12 | Rigel Pharmaceuticals, Inc. | Exocytosis pathway proteins and methods of use |
| AU3391200A (en) | 1999-03-04 | 2000-09-21 | Corixa Corporation | Compositions and methods for breast cancer therapy and diagnosis |
| EP2270148A3 (en) | 1999-04-09 | 2011-06-08 | Kyowa Hakko Kirin Co., Ltd. | Method for controlling the activity of immunologically functional molecule |
| US7534866B2 (en) | 2005-10-19 | 2009-05-19 | Ibc Pharmaceuticals, Inc. | Methods and compositions for generating bioactive assemblies of increased complexity and uses |
| EP1396543A3 (en) | 1999-07-08 | 2004-03-31 | Research Association for Biotechnology | Primers for synthesizing full length cDNA clones and their use |
| DE19932688B4 (de) | 1999-07-13 | 2009-10-08 | Scil Proteins Gmbh | Design von Beta-Faltblatt-Proteinen des gamma-II-kristallins antikörperähnlichen |
| ES2327382T3 (es) | 1999-07-20 | 2009-10-29 | Morphosys Ag | Metodos para presentar (poli)peptidos/proteinas en particulas de bacteriofagos a traves de enlaces disulfuro. |
| KR100942863B1 (ko) | 1999-08-24 | 2010-02-17 | 메다렉스, 인코포레이티드 | 인간 씨티엘에이-4 항체 및 그의 용도 |
| US20030138804A1 (en) | 2000-02-03 | 2003-07-24 | Boyle Bryan J. | Methods and materials relating to novel C-type lectin receptor-like polypeptides and polynucleotides |
| CA2405912A1 (en) | 2000-04-12 | 2001-10-18 | Human Genome Sciences, Inc. | Albumin fusion proteins |
| US7943129B2 (en) | 2000-05-26 | 2011-05-17 | National Research Council Of Canada | Single-domain brain-targeting antibody fragments derived from llama antibodies |
| ES2295228T3 (es) | 2000-11-30 | 2008-04-16 | Medarex, Inc. | Roedores transcromosomicos transgenicos para la preparacion de anticuerpos humanos. |
| CN1294148C (zh) | 2001-04-11 | 2007-01-10 | 中国科学院遗传与发育生物学研究所 | 环状单链三特异抗体 |
| US20050048512A1 (en) | 2001-04-26 | 2005-03-03 | Avidia Research Institute | Combinatorial libraries of monomer domains |
| US20050053973A1 (en) | 2001-04-26 | 2005-03-10 | Avidia Research Institute | Novel proteins with targeted binding |
| US20040175756A1 (en) | 2001-04-26 | 2004-09-09 | Avidia Research Institute | Methods for using combinatorial libraries of monomer domains |
| US7514594B2 (en) | 2001-05-11 | 2009-04-07 | Wyeth | Transgenic animal model of bone mass modulation |
| WO2002092000A2 (en) | 2001-05-11 | 2002-11-21 | Genome Therapeutics Corporation | Hbm variants that modulate bone mass and lipid levels |
| US20040038860A1 (en) | 2002-05-17 | 2004-02-26 | Allen Kristina M. | Reagents and methods for modulating dkk-mediated interactions |
| WO2002092780A2 (en) | 2001-05-17 | 2002-11-21 | Diversa Corporation | Novel antigen binding molecules for therapeutic, diagnostic, prophylactic, enzymatic, industrial, and agricultural applications, and methods for generating and screening thereof |
| CA2446582A1 (en) | 2001-05-17 | 2002-11-21 | Genome Therapeutics Corporation | Reagents and methods for modulating dkk-mediated interactions |
| AU2002319402B2 (en) | 2001-06-28 | 2008-09-11 | Domantis Limited | Dual-specific ligand and its use |
| ITMI20011483A1 (it) | 2001-07-11 | 2003-01-11 | Res & Innovation Soc Coop A R | Uso di composti come antagonisti funzionali ai recettori centrali deicannabinoidi |
| WO2003029469A1 (fr) | 2001-09-13 | 2003-04-10 | Juridical Foundation The Chemo-Sero-Therapeutic Research Institute | Nouvelles proteines contenant de la selenocysteine |
| HUP0600342A3 (en) | 2001-10-25 | 2011-03-28 | Genentech Inc | Glycoprotein compositions |
| US7335478B2 (en) | 2002-04-18 | 2008-02-26 | Kalobios Pharmaceuticals, Inc. | Reactivation-based molecular interaction sensors |
| US20030157579A1 (en) | 2002-02-14 | 2003-08-21 | Kalobios, Inc. | Molecular sensors activated by disinhibition |
| AU2003217912A1 (en) | 2002-03-01 | 2003-09-16 | Xencor | Antibody optimization |
| US20030194708A1 (en) | 2002-04-10 | 2003-10-16 | Minke Binnerts | Human homolog of crossveinless materials and methods |
| JP2006512895A (ja) | 2002-06-28 | 2006-04-20 | ドマンティス リミテッド | リガンド |
| EP1900753B1 (en) | 2002-11-08 | 2017-08-09 | Ablynx N.V. | Method of administering therapeutic polypeptides, and polypeptides therefor |
| AU2003290330A1 (en) | 2002-12-27 | 2004-07-22 | Domantis Limited | Dual specific single domain antibodies specific for a ligand and for the receptor of the ligand |
| US20050008625A1 (en) | 2003-02-13 | 2005-01-13 | Kalobios, Inc. | Antibody affinity engineering by serial epitope-guided complementarity replacement |
| WO2004100301A1 (ja) | 2003-05-07 | 2004-11-18 | Gs Yuasa Corporation | 直接形燃料電池システム |
| US20090005257A1 (en) | 2003-05-14 | 2009-01-01 | Jespers Laurent S | Process for Recovering Polypeptides that Unfold Reversibly from a Polypeptide Repertoire |
| US20050084494A1 (en) | 2003-05-21 | 2005-04-21 | Darwin Prockop | Inhibitors of Dkk-1 |
| DE10324447A1 (de) | 2003-05-28 | 2004-12-30 | Scil Proteins Gmbh | Generierung künstlicher Bindungsproteine auf der Grundlage von Ubiquitin |
| PL1639011T3 (pl) | 2003-06-30 | 2009-05-29 | Domantis Ltd | Pegilowane przeciwciała jednodomenowe (dAb) |
| US20050100543A1 (en) | 2003-07-01 | 2005-05-12 | Immunomedics, Inc. | Multivalent carriers of bi-specific antibodies |
| US9046537B2 (en) | 2003-09-22 | 2015-06-02 | Enzo Biochem, Inc. | Method for treating inflammation by administering a compound which binds LDL-receptor-related protein (LRP) ligand binding domain |
| US8637506B2 (en) | 2003-09-22 | 2014-01-28 | Enzo Biochem, Inc. | Compositions and methods for bone formation and remodeling |
| WO2005048913A2 (en) | 2003-11-24 | 2005-06-02 | Gunnar Westin | Mutant lrp5/6 wnt-signaling receptors in cancer diagnosis, prognosis and treatment |
| CA2553692C (en) | 2004-01-20 | 2014-10-07 | Kalobios, Inc. | Antibody specificity transfer using minimal essential binding determinants |
| US20060094046A1 (en) | 2004-02-11 | 2006-05-04 | Arie Abo | Compositions and methods relating to angiogenesis and tumorigenesis |
| US20060127919A1 (en) | 2004-02-25 | 2006-06-15 | Arie Abo | Compositions and methods relating to cell adhesion molecule L1 |
| CN100376599C (zh) | 2004-04-01 | 2008-03-26 | 北京安波特基因工程技术有限公司 | 基因工程重组抗cea抗cd3抗cd28线性单链三特异抗体 |
| US7684505B2 (en) | 2004-04-26 | 2010-03-23 | Qualcomm Incorporated | Method and apparatus for encoding interleaving and mapping data to facilitate GBPS data rates in wireless systems |
| CA2566498A1 (en) | 2004-05-14 | 2005-12-08 | The Regents Of The University Of California | Methods for treating cancer using anti-wnt2 monoclonal antibodies and sirna |
| US20060008844A1 (en) | 2004-06-17 | 2006-01-12 | Avidia Research Institute | c-Met kinase binding proteins |
| JP2008512352A (ja) | 2004-07-17 | 2008-04-24 | イムクローン システムズ インコーポレイティド | 新規な四価の二重特異性抗体 |
| AU2005267722B2 (en) | 2004-08-04 | 2009-10-08 | Amgen Inc. | Antibodies to Dkk-1 |
| CN101065151B (zh) | 2004-09-23 | 2014-12-10 | 健泰科生物技术公司 | 半胱氨酸改造的抗体和偶联物 |
| WO2006055635A2 (en) | 2004-11-15 | 2006-05-26 | Mount Sinai School Of Medicine Of New York University | Compositions and methods for altering wnt autocrine signaling |
| JP2008528010A (ja) | 2005-01-31 | 2008-07-31 | アブリンクス ナームローゼ フェンノートシャップ | 重鎖抗体の可変ドメイン配列を作出する方法 |
| US7838252B2 (en) | 2005-02-17 | 2010-11-23 | The Board Of Trustees Of The Leland Stanford Junior University | Methods and compositions for treating a subject having an anthrax toxin mediated condition |
| WO2007024715A2 (en) | 2005-08-19 | 2007-03-01 | Abbott Laboratories | Dual variable domain immunoglobin and uses thereof |
| KR20080080482A (ko) | 2005-09-07 | 2008-09-04 | 메디뮨 엘엘씨 | 독소가 컨쥬게이트된 eph 수용체 항체 |
| US10155816B2 (en) | 2005-11-28 | 2018-12-18 | Genmab A/S | Recombinant monovalent antibodies and methods for production thereof |
| US20070248628A1 (en) | 2005-12-06 | 2007-10-25 | Keller Lorraine H | Immunogens in cancer stem cells |
| CA2646508A1 (en) | 2006-03-17 | 2007-09-27 | Biogen Idec Ma Inc. | Stabilized polypeptide compositions |
| MX2008014722A (es) | 2006-05-19 | 2011-04-15 | Teva Pharma | Proteinas de fusion, usos de las mismas y procesos para producir las mismas. |
| WO2007142987A2 (en) | 2006-05-30 | 2007-12-13 | Wisconsin Alumni Research Foundation | Mammary stem cell marker |
| US20100129928A1 (en) | 2006-07-27 | 2010-05-27 | Roberto Polakewicz | Tyrosine Phosphorylation Sites |
| WO2008027739A2 (en) | 2006-08-28 | 2008-03-06 | Nuvelo, Inc. | Antibodies to ntb-a |
| US8198236B2 (en) * | 2007-04-30 | 2012-06-12 | Washington University | Methods and compositions for the treatment of cancer |
| JP2010527936A (ja) | 2007-05-18 | 2010-08-19 | メディミューン,エルエルシー | 炎症性疾患におけるil−33 |
| US20090176654A1 (en) | 2007-08-10 | 2009-07-09 | Protelix, Inc. | Universal fibronectin type III binding-domain libraries |
| CN101842116A (zh) | 2007-08-28 | 2010-09-22 | 比奥根艾迪克Ma公司 | 结合igf-1r多个表位的组合物 |
| US20090155255A1 (en) | 2007-09-27 | 2009-06-18 | Biogen Idec Ma Inc. | Cd23 binding molecules and methods of use thereof |
| AR068767A1 (es) | 2007-10-12 | 2009-12-02 | Novartis Ag | Anticuerpos contra esclerostina, composiciones y metodos de uso de estos anticuerpos para tratar un trastorno patologico mediado por esclerostina |
| WO2009051957A2 (en) | 2007-10-16 | 2009-04-23 | Nuvelo, Inc. | Antibodies to irem-1 |
| EP2211903A4 (en) | 2007-10-17 | 2011-07-06 | Nuvelo Inc | CLL-1 ANTIBODY |
| PL2567709T3 (pl) | 2007-11-02 | 2018-06-29 | Novartis Ag | Cząsteczki i sposoby modulowania białka związanego z receptorem dla lipoproteiny o niskiej gęstości 6 (LRP6) |
| MX2010005282A (es) | 2007-11-13 | 2010-08-31 | Teva Pharma | Anticuerpos humanizados contra tl1a. |
| CA2705923A1 (en) | 2007-11-16 | 2009-05-22 | Nuvelo, Inc. | Antibodies to lrp6 |
| EP2080812A1 (en) | 2008-01-18 | 2009-07-22 | Transmedi SA | Compositions and methods of detecting post-stop peptides |
| WO2009126920A2 (en) | 2008-04-11 | 2009-10-15 | Merrimack Pharmaceuticals, Inc. | Human serum albumin linkers and conjugates thereof |
| US8716243B2 (en) | 2008-05-28 | 2014-05-06 | St. Jude Childen's Research Hospital | Methods of effecting Wnt signaling through Dkk structural analysis |
| WO2009148896A2 (en) | 2008-05-29 | 2009-12-10 | Nuclea Biotechnologies, LLC | Anti-phospho-akt antibodies |
| TWI388570B (zh) | 2008-07-23 | 2013-03-11 | Hanmi Science Co Ltd | 包含具有三個官能端的非肽醯聚合物的多肽複合物 |
| KR20110112301A (ko) | 2008-11-18 | 2011-10-12 | 메리맥 파마슈티컬즈, 인크. | 인간 혈청 알부민 링커 및 그 콘쥬게이트 |
| JP5693475B2 (ja) | 2009-03-04 | 2015-04-01 | ペイタント・ソリューションズ・インコーポレイテッドPeytant Solutions, Inc. | 羊膜組織を含む材料で修飾されたステント及び対応する方法 |
| RU2542394C2 (ru) | 2009-03-24 | 2015-02-20 | ТЕВА БИОФАРМАСЬЮТИКАЛЗ ЮЭсЭй, ИНК. | Гуманизированные антитела против light и их применение |
| EP2419120A4 (en) | 2009-04-08 | 2016-01-06 | Univ California | HUMAN PROTEIN CONCERNS WITH CONTROLLED SERUM PHARMACOKINETICS |
| CN102459346B (zh) | 2009-04-27 | 2016-10-26 | 昂考梅德药品有限公司 | 制造异源多聚体分子的方法 |
| US20110023351A1 (en) | 2009-07-31 | 2011-02-03 | Exxonmobil Research And Engineering Company | Biodiesel and biodiesel blend fuels |
| CA2772929A1 (en) | 2009-09-03 | 2011-03-11 | Genentech, Inc. | Methods for treating, diagnosing, and monitoring rheumatoid arthritis |
| UY32920A (es) | 2009-10-02 | 2011-04-29 | Boehringer Ingelheim Int | Moleculas de unión biespecíficas para la terapia anti-angiogénesis |
| EP2488199A1 (en) | 2009-10-14 | 2012-08-22 | Merrimack Pharmaceuticals, Inc. | Bispecific binding agents targeting igf-1r and erbb3 signalling and uses thereof |
| WO2011084714A2 (en) | 2009-12-17 | 2011-07-14 | Biogen Idec Ma Inc. | STABILIZED ANTI-TNF-ALPHA scFv MOLECULES OR ANTI-TWEAK scFv MOLECULES AND USES THEREOF |
| WO2011103426A2 (en) | 2010-02-19 | 2011-08-25 | The Board Of Regents Of The University Of Oklahoma | Monoclonal antibodies that inhibit the wnt signaling pathway and methods of production and use thereof |
| WO2011106707A2 (en) | 2010-02-26 | 2011-09-01 | Human Genome Sciences, Inc. | Antibodies that specifically bind to dr3 |
| NZ602040A (en) | 2010-03-24 | 2014-12-24 | Genentech Inc | Anti-lrp6 antibodies |
| AU2011249782B2 (en) | 2010-05-06 | 2014-10-02 | Novartis Ag | Compositions and methods of use for therapeutic low density lipoprotein - related protein 6 (LRP6) multivalent antibodies |
| ES2659406T3 (es) | 2010-05-06 | 2018-03-15 | Novartis Ag | Composiciones y procedimientos de uso para anticuerpos terapéuticos contra la proteína 6 relacionada con las lipoproteínas de baja densidad (LRP6) |
| WO2012054565A1 (en) | 2010-10-20 | 2012-04-26 | Genentech, Inc. | Methods and compositions for modulating the wnt pathway |
| US20140017264A1 (en) | 2010-12-10 | 2014-01-16 | Merrimack Pharmaceuticals, Inc. | Dosage and administration of bispecific scfv conjugates |
| IL229503B (en) | 2011-06-23 | 2022-06-01 | Ablynx Nv | Techniques for predicting, detecting and reducing interference of a-specific protein in tests involving single variable domains of immunoglobulin |
| EP2773669B1 (en) | 2011-11-04 | 2018-03-28 | Novartis AG | Low density lipoprotein-related protein 6 (lrp6) - half life extender constructs |
-
2011
- 2011-05-05 ES ES11718982.9T patent/ES2659406T3/es active Active
- 2011-05-05 MX MX2012012928A patent/MX2012012928A/es not_active Application Discontinuation
- 2011-05-05 PE PE2012002125A patent/PE20130207A1/es not_active Application Discontinuation
- 2011-05-05 BR BR112012028306-5A patent/BR112012028306A2/pt not_active Application Discontinuation
- 2011-05-05 NZ NZ60382911A patent/NZ603829A/en unknown
- 2011-05-05 EP EP17205208.6A patent/EP3345926B1/en active Active
- 2011-05-05 CN CN201180035808.8A patent/CN103038258B/zh active Active
- 2011-05-05 EP EP23166606.6A patent/EP4234698A3/en active Pending
- 2011-05-05 KR KR1020127031870A patent/KR20130066632A/ko not_active Application Discontinuation
- 2011-05-05 CA CA2798432A patent/CA2798432A1/en active Pending
- 2011-05-05 EA EA201291180A patent/EA201291180A1/ru unknown
- 2011-05-05 ES ES17205208T patent/ES2949159T3/es active Active
- 2011-05-05 SG SG2012081188A patent/SG185416A1/en unknown
- 2011-05-05 EP EP11718982.9A patent/EP2566892B1/en active Active
- 2011-05-05 AU AU2011249783A patent/AU2011249783B9/en active Active
- 2011-05-05 US US13/696,532 patent/US9428583B2/en active Active
- 2011-05-05 WO PCT/EP2011/057202 patent/WO2011138392A1/en active Application Filing
- 2011-05-05 JP JP2013508501A patent/JP2013527762A/ja not_active Withdrawn
-
2012
- 2012-10-25 TN TNP2012000511A patent/TN2012000511A1/en unknown
- 2012-11-01 IL IL222811A patent/IL222811A0/en unknown
- 2012-11-01 ZA ZA2012/08221A patent/ZA201208221B/en unknown
- 2012-11-06 CL CL2012003094A patent/CL2012003094A1/es unknown
- 2012-11-06 CO CO12199736A patent/CO6630181A2/es unknown
- 2012-11-06 GT GT201200297A patent/GT201200297A/es unknown
-
2014
- 2014-01-23 CL CL2014000177A patent/CL2014000177A1/es unknown
- 2014-09-15 AU AU2014224144A patent/AU2014224144A1/en not_active Abandoned
-
2016
- 2016-03-17 US US15/073,141 patent/US20170362326A1/en not_active Abandoned
- 2016-07-14 JP JP2016139194A patent/JP2017008060A/ja not_active Withdrawn
-
2017
- 2017-11-30 JP JP2017230243A patent/JP2018065843A/ja not_active Withdrawn
-
2019
- 2019-01-10 HK HK19100400.3A patent/HK1258029A1/zh unknown
-
2020
- 2020-02-24 US US16/799,509 patent/US20200377603A1/en not_active Abandoned
- 2020-02-28 JP JP2020033213A patent/JP2020114210A/ja not_active Withdrawn
-
2022
- 2022-06-22 JP JP2022100111A patent/JP2022137092A/ja active Pending
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| KR20130066631A (ko) | 치료적 저밀도 지단백질-관련 단백질 6 (lrp6) 다가 항체에 대한 조성물 및 사용 방법 | |
| KR20130066632A (ko) | 치료적 저밀도 지단백질-관련 단백질 6 (lrp6) 항체에 대한 조성물 및 사용 방법 | |
| CN111434692B (zh) | 抗cld18a2纳米抗体及其应用 | |
| KR102161460B1 (ko) | St2 항원 결합 단백질 | |
| KR102105776B1 (ko) | Csf1r에 결합하는 항체들 | |
| KR20140091031A (ko) | 저밀도 지단백질-관련 단백질 6 (lrp6) - 반감기 연장제 구축물 | |
| KR20180081532A (ko) | 암 치료용 조성물 및 방법 | |
| KR20150023811A (ko) | 암의 치료를 위한 lsr 항체 및 그의 용도 | |
| KR20180054837A (ko) | 인간 cd40에 특이적으로 결합하는 작용성 항체 및 사용 방법 | |
| CN107206072A (zh) | T细胞活化性双特异性抗原结合分子CD3 ABD叶酸受体1(FolR1)和PD‑1轴结合拮抗剂的组合疗法 | |
| KR20140029446A (ko) | 면역 관련 장애 및 암의 치료를 위한 폴리펩티드 및 폴리뉴클레오티드, 및 이들의 용도 | |
| KR20150122761A (ko) | T 세포 활성화 항원 결합 분자 | |
| AU2011249782A1 (en) | Compositions and methods of use for therapeutic low density lipoprotein - related protein 6 (LRP6) multivalent antibodies | |
| KR20200003367A (ko) | 암 치료용 조성물 및 방법 | |
| KR20160021849A (ko) | 렉틴-유사 산화된 ldl 수용체1 항체 및 사용 방법 | |
| CN111954680A (zh) | IL2Rβ/共同γ链抗体 | |
| KR20230137393A (ko) | Psma 결합 단백질 및 이의 용도 | |
| KR20230059789A (ko) | 항-가변 muc1* 항체 및 이의 용도 | |
| Class et al. | Patent application title: Broadly Neutralizing Monoclonal Antibodies Against HIV-1 V1V2 Env Region | |
| CN117858904A (zh) | 特异性结合cd47和pd-l1的分离的双特异性抗体 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| WITN | Application deemed withdrawn, e.g. because no request for examination was filed or no examination fee was paid |