KR20200028039A - 3-에피메라아제 - Google Patents
3-에피메라아제 Download PDFInfo
- Publication number
- KR20200028039A KR20200028039A KR1020207006415A KR20207006415A KR20200028039A KR 20200028039 A KR20200028039 A KR 20200028039A KR 1020207006415 A KR1020207006415 A KR 1020207006415A KR 20207006415 A KR20207006415 A KR 20207006415A KR 20200028039 A KR20200028039 A KR 20200028039A
- Authority
- KR
- South Korea
- Prior art keywords
- sequence
- protein
- seq
- glu
- gly
- Prior art date
Links
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/90—Isomerases (5.)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P19/00—Preparation of compounds containing saccharide radicals
- C12P19/02—Monosaccharides
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P19/00—Preparation of compounds containing saccharide radicals
- C12P19/24—Preparation of compounds containing saccharide radicals produced by the action of an isomerase, e.g. fructose
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y501/00—Racemaces and epimerases (5.1)
- C12Y501/03—Racemaces and epimerases (5.1) acting on carbohydrates and derivatives (5.1.3)
-
- Y—GENERAL TAGGING OF NEW TECHNOLOGICAL DEVELOPMENTS; GENERAL TAGGING OF CROSS-SECTIONAL TECHNOLOGIES SPANNING OVER SEVERAL SECTIONS OF THE IPC; TECHNICAL SUBJECTS COVERED BY FORMER USPC CROSS-REFERENCE ART COLLECTIONS [XRACs] AND DIGESTS
- Y02—TECHNOLOGIES OR APPLICATIONS FOR MITIGATION OR ADAPTATION AGAINST CLIMATE CHANGE
- Y02P—CLIMATE CHANGE MITIGATION TECHNOLOGIES IN THE PRODUCTION OR PROCESSING OF GOODS
- Y02P20/00—Technologies relating to chemical industry
- Y02P20/50—Improvements relating to the production of bulk chemicals
- Y02P20/52—Improvements relating to the production of bulk chemicals using catalysts, e.g. selective catalysts
Landscapes
- Chemical & Material Sciences (AREA)
- Organic Chemistry (AREA)
- Life Sciences & Earth Sciences (AREA)
- Engineering & Computer Science (AREA)
- Zoology (AREA)
- Wood Science & Technology (AREA)
- Health & Medical Sciences (AREA)
- Genetics & Genomics (AREA)
- Bioinformatics & Cheminformatics (AREA)
- General Health & Medical Sciences (AREA)
- Biochemistry (AREA)
- General Engineering & Computer Science (AREA)
- Biotechnology (AREA)
- Microbiology (AREA)
- Chemical Kinetics & Catalysis (AREA)
- General Chemical & Material Sciences (AREA)
- Molecular Biology (AREA)
- Medicinal Chemistry (AREA)
- Biomedical Technology (AREA)
- Enzymes And Modification Thereof (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Preparation Of Compounds By Using Micro-Organisms (AREA)
- Peptides Or Proteins (AREA)
Abstract
SEQ ID NO: 6, SEQ ID NO: 2 또는 SEQ ID NO: 4에 대하여 적어도 70%의 서열 동일성을 가지는 폴리펩타이드 서열을 포함하는 단백질을 제공한다. 이 단백질은, 케토오스-3-에피메라아제 활성을 가진다.
Description
본 발명은, 케토오스-3-에피메라아제(ketose-3-epimerase) 활성을 가지는 단백질과, 상기 단백질을 인코드하는 핵산분자에 관한 것이다. 본 발명은 또한, 벡터와, 핵산분자를 가지는 숙주세포에 관한 것이다. 본 발명은 또한, 상기 단백질을 이용하여 알룰로오스(Allulose)를 합성하는 방법과, 그와 같이 하여 생성되는 알룰로오스에 관한 것이다.
알룰로오스는, "제로 칼로리"의 감미료이며, 덱스트로오스와 유사한 것으로서 제안되는 감미를 가진다. 알룰로오스도 또한, 다른 당류와 유사하게, 팽화 및 갈색화 등의 성질을 가진다. 알룰로오스의 주요한 타겟 시장은, 현재 제품에 덱스트로오스, 과당(fructose) 또는 HFCS를 사용하고 있고, 예를 들면 팽화, 갈색화, 질감, 및 감미 등의 당분으로 유발되는 성질을 현저히 바꾸지 않고, 칼로리를 현저히 저감시키는 것을 목표로 하고 있는 식품 제조사 및 음료 제조사이다.
알룰로오스는 통상, 미국에서는 일반적으로 안전하다고 인정되는 식품(Generally recognized as safe)은 아니지만, 현재 GRAS 통지가 보류 중이다(GRN400). 알룰로오스는, 가공한 사탕수수 및 사탕무 당밀, 수증기 처리한 커피, 밀 제품, 및 고과당 시럽 등에 존재한다. 알룰로오스의 일반적인 일상 총 섭취량은, 하루 0.2g을 상회한다고 추정되고 있다. D-알룰로오스는, D-과당의 C-3 에피머(epimer)이며, 알룰로오스와 과당의 구조상의 차이로 인해, 알룰로오스는 인체 내에서 대사되지 않기 때문에, 제로 칼로리가 된다. 따라서 알룰로오스는, 칼로리가 없는 데다가, 일반적인 단당류와 유사한 성질을 유지하면서, 감미가 있다고 보고되어 있기 때문에, 팽화성의 감미제로서 유망한 후보라고 여겨지고 있다.
본 발명은, 종래의 기술을 뛰어 넘기 위하여, 알룰로오스 생성의 개선을 목표로 하는 것이다. 본 발명은, 과거에 보고되었던 것보다도, 세포계 전체에 있어서의 전환율 및 용적 생산성이 높은 케토오스-3-에피메라아제를 제공하는 것을 목표로 하는 것이다.
본 발명은, 3개의 케토오스-3-에피메라아제 효소의 동정과 특징화에 기인하며, 그 일례로서의 아미노산 서열이 SEQ ID NO: 2, 4, 및 6에 나타나 있다. 이 케토오스-3-에피메라아제는, 과당을 알룰로오스로 전환하는 데 사용되어도 된다. 이들 단백질은, 종래에, 가설(hypothetical) 단백질로서 또는, 타가토오스 에피메라아제 활성을 가지는 것으로서 동정되어 왔다. 그러나 본 발명자는, 이들 효소가 사이코스-3-에피메라아제 활성을 가진다는 놀랄 만한 사실을 발견했다.
본 발명의 제1 양태에 의하면, 단백질로서, SEQ ID NO: 2, SEQ ID NO: 4 또는 SEQ ID NO: 6에 대하여 적어도 70%의 서열 동일성(identity)을 가지는 폴리펩타이드 서열을 포함하고, 상기 단백질은, 케토오스-3-에피메라아제 활성을 가지는 단백질을 제공한다.
폴리펩타이드 서열은, SEQ ID NO: 2, SEQ ID NO: 4 또는 SEQ ID NO: 6에 대하여 적어도 80%, 90%, 95% 또는 99%의 서열 동일성을 가지거나 혹은 SEQ ID NO: 2, SEQ ID NO: 4 또는 SEQ ID NO: 6에 대하여 100%의 서열 동일성을 가지는 것이 적합하다.
상기 폴리펩타이드 서열은, SEQ ID NO: 13의 서열을 포함하는 것이 바람직하다.
상기 단백질은, 고체기질 상에 고정화되는 것이 적합하다.
본 발명의 제2 양태에 의하면, 알룰로오스 합성을 위한, 본 발명의 제1 양태에 관한 단백질의 사용을 제공한다.
본 발명의 제3 양태에 의하면, 핵산분자로서, 본 발명의 제1 양태에 관한 단백질을 인코드하는 폴리뉴클레오타이드 서열을 포함하는 핵산분자를 제공한다.
상기 핵산분자는,
i) SEQ ID NO: 5, SEQ ID NO: 1 또는 SEQ ID NO: 3에 대하여 적어도 70%, 80%, 90%, 95% 또는 99%의 서열 동일성을 가지거나 혹은 SEQ ID NO: 5, SEQ ID NO: 1 또는 SEQ ID NO: 3에 대하여 100%의 서열 동일성을 가지고,
ii) 엄격한(stringent) 조건하에 있어서, SEQ ID NO: 5, SEQ ID NO: 1 또는 SEQ ID NO: 3에 규정된 서열에 대하여 상보적인 서열을 가지는 폴리뉴클레오타이드에 혼성화하는
폴리뉴클레오타이드 서열을 포함하는 것이 유리하다.
본 발명의 제4 양태에 의하면, 본 발명의 제3 양태에 관한 핵산분자를 포함하는 벡터를 제공한다.
본 발명의 제5 양태에 의하면, 본 발명의 제3 양태에 관한 재조합 핵산분자를 포함하는 숙주세포를 제공한다.
상기 숙주세포는 효모, 박테리아 또는 그 이외의 미생물이거나 혹은 포유동물, 식물 또는 그 이외의 세포 배양물인 것이 적합하다.
상기 숙주세포는 대장균인 것이 적합하다.
본 발명의 제6 양태에 의하면, 본 발명의 제1 양태에 관한 단백질에 의하여 생성되는 알룰로오스를 제공한다.
본 발명의 제7 양태에 의하면, 알룰로오스의 생성방법으로서,
i) 본 발명의 제1 양태에 관한 단백질을 제공하는 단계와,
ii) 과당 기질을 알룰로오스로 전환하는 조건하에 있어서, 상기 단백질을 상기 과당 기질에 접촉시키는 단계를 포함하는 방법을 제공한다. 알룰로오스의 생성방법도 본 발명에 의하여 제공하며, 이 방법은 과당 기질을 알룰로오스로 전환하는 조건하에 있어서, 본 발명의 제1 양태에 관한 단백질을 상기 과당 기질에 접촉시키는 단계를 포함한다.
상기 단백질은, 숙주세포 내에 존재하는 것이 유리하다.
혹은 상기 단백질은 단리(isolated) 형태이다.
상기 조건에는 상기 단백질 및 상기 과당 기질을 25℃~75℃, 바람직하게는 50℃~60℃, 더욱 바람직하게는 52℃~55℃의 온도 범위, 더욱 바람직하게는 55℃로 유지하는 것이 포함되는 것이 적합하다.
상기 조건에는, 상기 단백질 및 상기 과당 기질을 pH4~pH10의 범위로 유지하는 것이 포함되는 것이 적합하다.
상기 조건에는 상기 과당 기질을 75%~95%의 농도 범위로 유지하는 것이 포함되는 것이 유리하다.
본 발명의 제8 양태에 의하면, 핵산분자로서,
i) SEQ ID NO: 5, SEQ ID NO: 1 또는 SEQ ID NO: 3에 대하여 적어도 70%의 서열 동일성을 가지고,
ii) 엄격한 조건하에 있어서, SEQ ID NO: 5, SEQ ID NO: 1 또는 SEQ ID NO: 3에 규정된 서열에 대하여 상보적인 서열을 가지는 폴리뉴클레오타이드에 혼성화하는
폴리뉴클레오타이드 서열을 포함하는 핵산분자를 제공한다.
이 핵산분자는, 케토오스-3-에피메라아제 활성을 가지는 폴리펩타이드를 인코드한다. 본 발명의 일 양태에 의하면, 이 핵산분자는 단리 형태이어도 된다.
본 발명의 제9 양태에 의하면, 케토오스-3-에피메라아제 활성을 가지는 폴리펩타이드를 인코드하는 폴리뉴클레오타이드 서열을 포함한 재조합 핵산분자를 포함하는 숙주세포로서, 상기 폴리뉴클레오타이드 서열은,
i) SEQ ID NO: 5, SEQ ID NO: 1 또는 SEQ ID NO: 3에 대하여 적어도 70%의 서열 동일성을 가지고,
ii) 엄격한 조건하에 있어서, SEQ ID NO: 5, SEQ ID NO: 1 또는 SEQ ID NO: 3에 규정된 서열에 대하여 상보적인 서열을 가지는 폴리뉴클레오타이드에 혼성화하는
숙주세포를 제공한다.
본 발명의 제10 양태에 의하면, 케토오스-3-에피메라아제 활성을 가지는 폴리펩타이드를 인코드하는 폴리뉴클레오타이드를 포함한 핵산분자를 포함하는 벡터로서, 상기 폴리뉴클레오타이드 서열은,
i) SEQ ID NO: 5, SEQ ID NO: 1 또는 SEQ ID NO: 3에 대하여 적어도 70%의 서열 동일성을 가지고,
ii) 엄격한 조건하에 있어서, SEQ ID NO: 5, SEQ ID NO: 1 또는 SEQ ID NO: 3에 규정된 서열에 대하여 상보적인 서열을 가지는 폴리뉴클레오타이드에 혼성화하는
벡터를 제공한다.
본 발명의 제11 양태에 의하면, 알룰로오스의 생성방법으로서,
i) 케토오스-3-에피메라아제 활성을 가지는 단백질을 인코드하는 폴리뉴클레오타이드 서열을 가지는 핵산분자를 포함한 벡터를 제공하는 단계를 포함하고, 상기 폴리뉴클레오타이드 서열은, a) SEQ ID NO: 5, SEQ ID NO: 1 또는 SEQ ID NO: 3에 대하여 적어도 70%의 서열 동일성을 가지며, b) 엄격한 조건하에 있어서, SEQ ID NO: 5, SEQ ID NO: 1 또는 SEQ ID NO: 3에 규정된 서열에 대하여 상보적인 서열을 가지는 폴리뉴클레오타이드에 혼성화하고, 상기 방법은 또한,
ii) 상기 폴리뉴클레오타이드 서열에 인코드된 케토오스-3-에피메라아제 활성을 가지는 상기 단백질을 합성하는 단계와,
iii) 과당을 케토오스-3-에피메라아제 활성을 가지는 상기 단백질에 접촉시켜, 과당으로부터 알룰로오스로의 전환을 허용하는 조건하에 있어서, 상기 과당 및 상기 단백질을 유지하는 단계와,
iv) 상기 단계 iii)에 있어서 생성된 상기 알룰로오스를 적어도 부분적으로 정제하는 단계를 포함하는 방법을 제공한다.
본 명세서에 있어서 "폴리펩타이드", "펩타이드" 및 "단백질"이라는 용어는, 아미노산 잔기의 폴리머를 가리키며 동일한 의미로 사용되고 있다. 이들 용어는 또한, 1개 이상의 아미노산 잔기가 수식 잔기이거나, 대응하는 천연 유래의 아미노산의 인공 화학적 모방체(mimetic) 등, 비천연 유래의 잔기인 아미노산 폴리머나, 천연 유래의 아미노산 폴리머에 적용된다. 폴리펩타이드는, "단리 형태"여도 되고 아니어도 되며, 즉, 천연 유래인 경우, 주위에 존재하는 성분으로부터 제거되어도 되고 그렇지 않아도 된다.
본 명세서에 있어서 사용하는 "아미노산"이라는 용어는, 천연 유래의 아미노산, 합성 아미노산, 및 천연 유래의 아미노산과 유사한 기능을 가지는 아미노산 유사체(analogues)나 아미노산 모방체를 가리킨다. 천연 유래의 아미노산은, 유전자 코드로 인코드된 것이나, 세포 내에서 번역된 후에 수식된(modified) 것(예를 들면, 하이드록시프롤린, γ-카르복시글루탐산, 및 O-포스포세린)이다. 이 "아미노산 유사체"라는 말은, 천연 유래의 아미노산과 동일한 기본적 화학구조(수소, 카르복시기, 아미노기, 및 R기에 결합하는 α탄소)를 가지지만, 수식된 R기 또는 수식된 골결(예를 들면, 호모세린, 노르류신, 메싸이오닌설폭사이드, 메싸이오닌메틸설포늄)을 가진다. "아미노산 모방체"라는 말은, 천연 유래의 아미노산과는 상이한 구조를 가지지만 유사한 기능을 구비한 화학 화합물을 가리킨다. 특정의 폴리펩타이드를 인코드하는 핵산분자는, 유전자 코드의 축퇴(degeneracy)에 의하여, 폴리뉴클레오타이드 서열의 범위를 가져도 된다는 것이 이해되어야 한다. 예를 들면, 코돈 GCA, GCC, GCG, 및 GCT는 모두, 아미노산 알라닌을 인코드한다.
2개의 서열의 백분율에 의한 "동일성"은, BLASTP 알고리즘 버젼 2.2.2(Altschul, Stephen F., Thomas L. Madden, Alejandro A. Schaffer, Jinghui Zhang, Zheng Zhang, Webb Miller, 및 David J. Lipman(1997), "갭 BLAST 및 PSI-BLAST: 단백질 데이터베이스 검색 프로그램의 신규 작성(Gapped BLAST and PSI-BLAST: a new generation of protein database search programs)", Nucleic Acids Res. 25: 3389-3402)를 사용하며, 디폴트 파라미터를 사용하여 판정해도 된다. 특히, BLAST 알고리즘에 대해서는, 인터넷 상에서 http: //www.ncbi.nlm.nih.gov/blast/의 URL에 액세스할 수 있다.
본 명세서에 있어서 사용하는 "케토오스-3-에피메라아제 활성"이라는 용어는, 케토오스의 입체화학의 반전, 특히 과당으로부터 알룰로오스로의 전환을 촉매할 수 있는 효소를 의미한다. 예를 들면 일 실시형태에 의하면, "케토오스-3-에피메라아제 활성"은, 과당으로부터 알룰로오스에 대한 상호 전환율을, 효소가 존재하지 않는 것과 동일한 조건하에 있어서, 반응 혼합물 전체에 걸쳐 첨가된 효소(0.1U/mg)의 mg당 적어도 10마이크로몰/분 정도 증가시키는 효소의 능력이라고 규정된다. 대체로서의 실시형태에 있어서, 적어도 0.05U/mg 또는 0.2U/mg의 과당으로부터 알룰로오스에 대한 상호 전환율의 증가가 "케토오스-3-에피메라아제 활성"이라고 생각된다. D-과당으로부터 알룰로오스로의 전환에 있어서의 효소의 활성을 판정하기 위한 적합한 어세이는 이하와 같다. 1ml의 D-과당(50g/L), 트리스-HCL 버퍼(50mM, pH8.0), 및 0.5μM의 효소를 함유하는 반응 혼합물을 55℃에서 2분간 배양한다. 이 반응을 비등에 의하여 10분 후에 정지한다. 생성된 D-알룰로오스의 양은 HPLC법에 의하여 판정한다. 효소 활성의 1단위를 pH8.0 및 55℃에 있어서 1분당 1μ몰의 D-알룰로오스 형성을 촉매하는 효소의 양으로서 규정한다(J. Agric. Food Chem. 2011, 59, 7785-7792).
본 명세서에 있어서 "유전자", "폴리뉴클레오타이드", 및 "핵산분자"라는 용어는, 복수의 뉴클레오타이드의 폴리머를 가리키며 동일한 의미로 사용한다. 핵산분자는, 천연 유래의 핵산(즉, DNA 또는 RNA)을 포함하거나 혹은 펩타이드 핵산, 모르폴린, 잠금(locked) 핵산 등의 인공 핵산이나, 글리콜 핵산 및 트레오스(threose) 핵산을 포함해도 된다.
본 명세서에서 사용되는 "뉴클레오타이드"라는 용어는, 천연 유래의 뉴클레오타이드와, 세포성 효소에 의하여 인식되는 합성 뉴클레오타이드 유사체를 가리킨다.
본 명세서에서 사용되는 "벡터"라는 용어는, 세포성 전사(transcription)의 대상이 될 수 있는 핵산분자 및/또는 번역효소를 함유한 임의의 천연 또는 인공의 구조물을 가리킨다. 일례로서의 벡터에는, 플라스미드, 바이러스(박테리오파지를 포함함), 코스미드(cosmid), 인공 염색체 또는 전이인자(transposable element)가 포함된다.
본 명세서에서 사용되는 "숙주세포"라는 용어는, 배지에서 배양할 수 있고, 또한, 재조합 유전자의 발현에 사용할 수 있는 임의의 생체세포를 가리킨다. 이와 같은 숙주세포는, 진핵성 또는 원핵성이어도 되고, 세균성 세포 등의 미생물이어도 되며 또는, 세포주 유래의 세포(포유동물의 불사의 세포주 등)여도 된다.
본 명세서에서 사용되는 "엄격한 조건"이라는 용어는, 혼성화 조건에 대하여 언급하는 경우, 65℃에서 적어도 약 6X의 SSC 및 1%의 SDS, 0.1X의 SSC에 있어서의 약 20%(v/v)의 포름알데하이드에 의하여 약 42℃에서 10분간의 최초의 세정, 또한, 65℃에서 0.2X의 SSC 및 0.1%의 SDS에 의하여 계속되는 세정을 행하는 것을 의미한다. 종래, 본 명세서에 기재된 바와 같은 엄격한 조건하에서 기지의 핵산을 프로브로서 사용하는 혼성화 기술은, 구조적으로 유사한 핵산을 동정하는 것이라고 알려져 있다.
본 명세서에서 사용되는 "알룰로오스"라는 용어는, 화학식 I에 나타나는 구조의 단당을 가리킨다. 이것은 "D-사이코스"로서도 알려져 있다.
[화학식 I]
본 명세서에서 사용되는 "과당"이라는 용어는, 화학식 II에 나타나는 구조를 가지는 단당을 가리킨다. 과당 기질의 예로서는, 결정 과당 및 결정 과당 그린(crystalline fructose greens)이 포함되지만, 이에 한정되는 것은 아니다. 본 명세서에서 사용되는, "결정 과당 그린"이란, 결정화 모액의 비결정화 부분으로부터의 과당 결정이 행해지는 동안에 생성되는 프로세스의 흐름을 가리킨다.
[화학식 II]
본 명세서에서 이용되는 "재조합"이라는 용어는, 비천연 유래의 문맥에 있어서, 인공적인 개입에 의하여 생성된 핵산분자 또는 폴리펩타이드를 가리킨다. 예를 들면, 다른 폴리펩타이드로부터 단리된 제1 폴리펩타이드 또는, 천연으로 제1 폴리펩타이드에 연결시킬 수 있는 임의의 폴리펩타이드와는 상이한 아미노산 서열을 가지는 제2 폴리펩타이드 서열과 펩타이드 결합에 의하여 결합되는 제1 폴리펩타이드가 재조합 폴리펩타이드이다.
도 1은, 본 발명의 일 실시형태에 관한 클로스트리듐신덴스로부터의 케토오스-3-에피메라아제의 아미노산 서열을 나타낸다(SEQ ID NO: 2).
도 2는, 본 발명의 다른 실시형태에 관한 클로스트리듐하이레몬아에(Clostridium hylemonae)로부터의 케토오스-3-에피메라아제의 아미노산 서열을 나타낸다(SEQ ID NO: 4).
도 3은, 본 발명의 또 다른 실시형태에 관한 데스모스포라종으로부터의 케토오스-3-에피메라아제의 아미노산 서열을 나타낸다(SEQ ID NO: 6).
도 4는, 클로스트리듐셀룰롤리티쿰(Clostridium cellulolyticum)으로부터의 종래 기지의 자일로오스아이소메라아제의 아미노산 서열을 나타낸다(SEQ ID NO: 8).
도 5는, 도 1~도 3에 나타내는 3개의 케토오스-3-에피메라아제의 사이의 서열 비교와, 종래 기지의 3개의 케토오스-3-에피메라아제의 서열 비교를 나타낸다. 완전 보존 잔기를 하이라이트로 나타낸다.
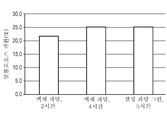
도 6은, 본 발명의 일 실시형태 및 대조군에 관한 효소를 발현하도록 형질전환된 대장균에 의한, 과당으로부터 알룰로오스로의 전환율을 나타내는 그래프이다.
도 7은, 도 4에 나타내는 아미노산 서열을 인코드하는 최적화 유전자 서열(SEQ ID NO: 7)과, 이 최적화 서열의 본래의 서열의 비교를 나타낸다.
도 8은, 도 3에 나타내는 아미노산 서열을 인코드하는 최적화 유전자 서열(SEQ ID NO: 5)과, 이 최적화 서열의 본래의 서열의 비교를 나타낸다.
도 9는, 도 1에 나타내는 아미노산 서열을 인코드하는 최적화 유전자 서열(SEQ ID NO: 3)과, 이 최적화 서열의 본래의 서열의 비교를 나타낸다.
도 10은, 도 2에 나타내는 아미노산 서열을 인코드하는 최적화 유전자 서열(SEQ ID NO: 3)과, 이 최적화 서열의 본래의 서열의 비교를 나타낸다.
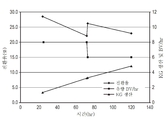
도 11은, 18L 스케일로 본 발명의 일 실시형태(데스모스포라종으로부터의 케토오스-3-에피메라아제)에 관한 효소를 발현하도록 형질전환되는 대장균에 의한 과당 기질로부터 알룰로오스로의 전환 준비를 나타내는 그래프이다.
도 12는, 본 발명의 실시형태(CH P3E, CS P3E, 및 DS P3E) 및 기지의 케토오스-3-에피메라아제(CC P3E)에 관한 효소에 의한 알룰로오스로의 전환율을 나타내는 그래프이다.
도 13은, A568 수지를 충전한 30ml의 고정상 반응기에 있어서의 DS P3E에 의한 알룰로오스의 전환율을 나타내는 그래프이다.
도 14는, A568 수지를 충전한 300ml의 고정상 반응기에 있어서의 DS P3E에 의한 알룰로오스의 전환율을 나타내는 그래프이다.
도 15는, 데스모스포라종으로부터의 천연 유래의 케토오스-3-에피메라아제의 인공 변이형의 아미노산 서열을 나타낸다(SEQ ID NO: 13).
서열 리스트의 간단한 설명
SEQ ID NO: 1은, 클로스트리듐신덴스로부터의 케토오스-3-에피메라아제를 인코드하는 유전자 서열(대장균에 있어서의 발현에 대하여 최적화)을 나타낸다.
SEQ ID NO: 2는, SEQ ID NO. 1의 유전자 서열에 의하여 인코드되는 케토오스-3-에피메라아제의 아미노산 서열을 나타낸다.
SEQ ID NO: 3은, 클로스트리듐하이레몬아에로부터의 케토오스-3-에피메라아제를 인코드하는 유전자 서열(대장균에 있어서의 발현에 대하여 최적화)을 나타낸다.
SEQ ID NO: 4는, SEQ ID NO. 3의 유전자 서열에 의하여 인코드되는 케토오스-3-에피메라아제의 아미노산 서열을 나타낸다.
SEQ ID NO: 5는, 데스모스포라 8437종으로부터의 케토오스-3-에피메라아제를 인코드하는 유전자 서열(대장균에 있어서의 발현에 대하여 최적화)을 나타낸다.
SEQ ID NO: 6은, SEQ ID NO. 5의 유전자 서열에 의하여 인코드되는 케토오스-3-에피메라아제의 아미노산 서열을 나타낸다.
SEQ ID NO: 7은, 클로스트리듐셀룰롤리티쿰으로부터의 케토오스-3-에피메라아제를 인코드하는 유전자 서열(대장균에 있어서의 발현에 대하여 최적화)을 나타낸다.
SEQ ID NO: 8은, SEQ ID NO. 7의 유전자 서열에 의하여 인코드되는 케토오스-3-에피메라아제의 아미노산 서열을 나타낸다.
SEQ ID NO: 9는, 클로스트리듐신덴스로부터의 케토오스-3-에피메라아제를 인코드하는 천연 유래의 유전자 서열을 나타낸다.
SEQ ID NO: 10은, 클로스트리듐하이레몬아에로부터의 케토오스-3-에피메라아제를 인코드하는 천연 유래의 유전자 서열을 나타낸다.
SEQ ID NO: 11은, 데스모스포라 8437종으로부터의 케토오스-3-에피메라아제를 인코드하는 천연 유래의 유전자 서열을 나타낸다.
SEQ ID NO: 12는, 클로스트리듐셀룰롤리티쿰으로부터의 케토오스-3-에피메라아제를 인코드하는 천연 유래의 유전자 서열을 나타낸다.
SEQ ID NO: 13은, 데스모스포라 8437종의 케토오스-3-에피메라아제의 인공 변이형의 아미노산 서열을 나타낸다.
도 2는, 본 발명의 다른 실시형태에 관한 클로스트리듐하이레몬아에(Clostridium hylemonae)로부터의 케토오스-3-에피메라아제의 아미노산 서열을 나타낸다(SEQ ID NO: 4).
도 3은, 본 발명의 또 다른 실시형태에 관한 데스모스포라종으로부터의 케토오스-3-에피메라아제의 아미노산 서열을 나타낸다(SEQ ID NO: 6).
도 4는, 클로스트리듐셀룰롤리티쿰(Clostridium cellulolyticum)으로부터의 종래 기지의 자일로오스아이소메라아제의 아미노산 서열을 나타낸다(SEQ ID NO: 8).
도 5는, 도 1~도 3에 나타내는 3개의 케토오스-3-에피메라아제의 사이의 서열 비교와, 종래 기지의 3개의 케토오스-3-에피메라아제의 서열 비교를 나타낸다. 완전 보존 잔기를 하이라이트로 나타낸다.
도 6은, 본 발명의 일 실시형태 및 대조군에 관한 효소를 발현하도록 형질전환된 대장균에 의한, 과당으로부터 알룰로오스로의 전환율을 나타내는 그래프이다.
도 7은, 도 4에 나타내는 아미노산 서열을 인코드하는 최적화 유전자 서열(SEQ ID NO: 7)과, 이 최적화 서열의 본래의 서열의 비교를 나타낸다.
도 8은, 도 3에 나타내는 아미노산 서열을 인코드하는 최적화 유전자 서열(SEQ ID NO: 5)과, 이 최적화 서열의 본래의 서열의 비교를 나타낸다.
도 9는, 도 1에 나타내는 아미노산 서열을 인코드하는 최적화 유전자 서열(SEQ ID NO: 3)과, 이 최적화 서열의 본래의 서열의 비교를 나타낸다.
도 10은, 도 2에 나타내는 아미노산 서열을 인코드하는 최적화 유전자 서열(SEQ ID NO: 3)과, 이 최적화 서열의 본래의 서열의 비교를 나타낸다.
도 11은, 18L 스케일로 본 발명의 일 실시형태(데스모스포라종으로부터의 케토오스-3-에피메라아제)에 관한 효소를 발현하도록 형질전환되는 대장균에 의한 과당 기질로부터 알룰로오스로의 전환 준비를 나타내는 그래프이다.
도 12는, 본 발명의 실시형태(CH P3E, CS P3E, 및 DS P3E) 및 기지의 케토오스-3-에피메라아제(CC P3E)에 관한 효소에 의한 알룰로오스로의 전환율을 나타내는 그래프이다.
도 13은, A568 수지를 충전한 30ml의 고정상 반응기에 있어서의 DS P3E에 의한 알룰로오스의 전환율을 나타내는 그래프이다.
도 14는, A568 수지를 충전한 300ml의 고정상 반응기에 있어서의 DS P3E에 의한 알룰로오스의 전환율을 나타내는 그래프이다.
도 15는, 데스모스포라종으로부터의 천연 유래의 케토오스-3-에피메라아제의 인공 변이형의 아미노산 서열을 나타낸다(SEQ ID NO: 13).
서열 리스트의 간단한 설명
SEQ ID NO: 1은, 클로스트리듐신덴스로부터의 케토오스-3-에피메라아제를 인코드하는 유전자 서열(대장균에 있어서의 발현에 대하여 최적화)을 나타낸다.
SEQ ID NO: 2는, SEQ ID NO. 1의 유전자 서열에 의하여 인코드되는 케토오스-3-에피메라아제의 아미노산 서열을 나타낸다.
SEQ ID NO: 3은, 클로스트리듐하이레몬아에로부터의 케토오스-3-에피메라아제를 인코드하는 유전자 서열(대장균에 있어서의 발현에 대하여 최적화)을 나타낸다.
SEQ ID NO: 4는, SEQ ID NO. 3의 유전자 서열에 의하여 인코드되는 케토오스-3-에피메라아제의 아미노산 서열을 나타낸다.
SEQ ID NO: 5는, 데스모스포라 8437종으로부터의 케토오스-3-에피메라아제를 인코드하는 유전자 서열(대장균에 있어서의 발현에 대하여 최적화)을 나타낸다.
SEQ ID NO: 6은, SEQ ID NO. 5의 유전자 서열에 의하여 인코드되는 케토오스-3-에피메라아제의 아미노산 서열을 나타낸다.
SEQ ID NO: 7은, 클로스트리듐셀룰롤리티쿰으로부터의 케토오스-3-에피메라아제를 인코드하는 유전자 서열(대장균에 있어서의 발현에 대하여 최적화)을 나타낸다.
SEQ ID NO: 8은, SEQ ID NO. 7의 유전자 서열에 의하여 인코드되는 케토오스-3-에피메라아제의 아미노산 서열을 나타낸다.
SEQ ID NO: 9는, 클로스트리듐신덴스로부터의 케토오스-3-에피메라아제를 인코드하는 천연 유래의 유전자 서열을 나타낸다.
SEQ ID NO: 10은, 클로스트리듐하이레몬아에로부터의 케토오스-3-에피메라아제를 인코드하는 천연 유래의 유전자 서열을 나타낸다.
SEQ ID NO: 11은, 데스모스포라 8437종으로부터의 케토오스-3-에피메라아제를 인코드하는 천연 유래의 유전자 서열을 나타낸다.
SEQ ID NO: 12는, 클로스트리듐셀룰롤리티쿰으로부터의 케토오스-3-에피메라아제를 인코드하는 천연 유래의 유전자 서열을 나타낸다.
SEQ ID NO: 13은, 데스모스포라 8437종의 케토오스-3-에피메라아제의 인공 변이형의 아미노산 서열을 나타낸다.
본 발명은, SEQ ID NO. 2, 4 또는 6에 나타내는 아미노산 서열을 가지는 폴리펩타이드를 구비한 단백질 전반에 관한 것이다. SEQ ID NO. 2, 4, 및 6의 폴리펩타이드의 소스 유기체(source organisms)를 표 1에 나타낸다.
| 소스 유기체 | SEQ ID NO. |
| 클로스트리듐신덴스 ATCC 35704 | 2 |
| 클로스트리듐하이레몬아에 DSM 15053 | 4 |
| 데스모스포라 8437종 | 6 |
그러나, 대체의 실시형태에 의하면, 폴리펩타이드 서열은, SEQ ID NO. 2, 4 또는 6에 나타내는 것과 동일하지는 않지만, 적어도 70%의 서열 동일성을 가진다. 폴리펩타이드 서열은, SEQ ID NO. 2, 4 또는 6에 대하여 적어도 80%, 90%, 95% 또는 99%의 서열 동일성을 가지거나, SEQ ID NO. 2, 4 또는 6에 대하여 100%의 서열 동일성을 가지는 것이 바람직하다.예를 들면, 일 실시형태에 있어서, 폴리펩타이드 서열은, SEQ ID NO. 6에 대하여 89%의 서열 동일성을 가지는 SEQ ID NO. 13의 서열을 포함한다. 폴리펩타이드 서열은, 케토오스-3-에피메라아제 활성을 가진다.
따라서 몇 가지의 실시형태에 있어서, 펩타이드의 1개 이상의 아미노산이 생략되거나 혹은 상이한 아미노산, 바람직하게는 유사한 아미노산으로 치환된다. 유사한 아미노산은, 관련 성질을 가지는 측쇄 부분을 가지는 아미노산이며, 천연 유래의 아미노산을 이하의 그룹으로 분류해도 된다. 라이신, 아르지닌, 히스티딘 등, 염기성 측쇄를 가지는 그룹. 아스파르트산 및 글루탐산 등, 산성 측쇄를 가지는 그룹. 아스파라진(aspargine), 글루타민, 세린, 트레오닌, 및 타이로신 등, 전하를 가지지 않는 극성 측쇄를 가지는 그룹. 글라이신, 알라닌, 발린, 류신, 아이소류신, 프롤린, 페닐알라닌, 메싸이오닌, 트립토판, 및 시스테인 등, 비극성 측쇄를 가지는 그룹. 따라서, 이들 그룹 내의 아미노산으로 치환하는 것이 바람직하다.
일반적으로, 폴리펩타이드는, (인비트로로 합성되어도 되지만)천연 유래의 폴리펩타이드의 화학적 성질과 일치하는 것이 바람직하지만, 몇 가지의 대체의 실시형태에 있어서, 폴리펩타이드는 펩타이드 모방체이다. 즉, 폴리펩타이드를 천연에서는 발생하지 않도록 수식한 것이다. 이와 같은 펩타이드 모방체에는, 천연 유래의 아미노산을 합성 아미노산으로 치환한 것, 및/또는, 폴리펩타이드 골격의 수식이 포함된다. 예를 들면 몇 가지의 실시형태에 있어서, 펩타이드 결합은, 레트로-인버소 펩타이드 모방체(Meziere 등에 의한 J Immunol, 1997년 10월, 1;159(7):3230-7 참조. 참조로서 본 명세서에 원용함)를 생성하기 위하여, 리버스 펩타이드 결합과 치환된다. 혹은, 아미노산은, 폴리머쇄를 형성하는 아미노산 잔기의 간격 및 배향을 유지하는 펩타이드 결합 이외의 공유결합에 의하여 연결되어 있다.
본 발명에 관한 이와 같은 수식 폴리펩타이드 및 비수식 폴리펩타이드는 모두, 케토오스-3-에피메라아제폴리메라아제 활성을 가진다. 즉, 단백질은, 정제되거나 숙주세포 내에 발현하면, 과당으로부터 알룰로오스로의 전환을 촉매하는 능력을 가진다. 케토오스-3-에피메라아제 활성을 가지는지 아닌지를 시험할 때의 적합한 조건을 실시예 1에 나타낸다.
본 발명에 관한 폴리펩타이드는, 전체 세포 내에 함유되어도 되고 혹은 단리 형태의 단백질, 부분적으로 정제된 단백질 또는 고정화 단백질이어도 된다. 단백질의 정제는, 세포 파괴 및 여과 등, 표준 방법에 따라 행해져도 된다. 다른 표준 방법은, 당업자에게 기지이다.
본 발명의 실시형태에 있어서, SEQ ID NO. 2, 4 또는 6과 적어도 70%의 서열 동일성을 가지는 아미노산 서열을 포함한 단백질을 인코드하는 폴리뉴클레오타이드 서열을 포함한 핵산분자를 제공한다. 여기에서 단백질은, 케토오스-3-에피메라아제 활성을 가진다. 예를 들면, 일 실시형태에 의하면, 핵산분자는, SEQ ID NO. 13의 폴리펩타이드 서열을 인코드하는 서열을 포함한다.
본 발명의 단백질을 특이적으로 인코드하는 서열에 더하여, 핵산분자는, 프라이머 영역, 전사인자 결합영역, 벡터 삽입영역 등의 다른 서열이나, 핵산 분해를 억제하는 서열(예를 들면, 폴리아데노신 신장부분)을 포함해도 된다. 핵산분자는, DNA 또는 RNA여도 되고, 본 발명의 단백질을 합성하기 위하여, 폴리뉴클레오타이드가 여전히 번역 가능한 경우, 합성 뉴클레오타이드를 포함해도 된다.
상기 서술한 바와 같이, 본 발명에 관한 단백질의 아미노산 서열은, 본 명세서에 개시된 특정의 서열과는 상이해도 된다. 바람직한 실시형태에 있어서, 핵산분자는, 대장균 숙주세포 내의 발현에 의하여 최적화된, SEQ ID NO. 1, 3 또는 5의 서열을 가지는 폴리뉴클레오타이드를 구비한다. 대체의 실시형태에 있어서, 폴리뉴클레오타이드 서열은, SEQ ID NO. 1, 3 또는 5 중 어느 1개와 적어도 70%의 서열 동일성을 가지고, 케토오스-3-에피메라아제 활성을 가지는 단백질을 인코드한다. 폴리뉴클레오타이드 서열은, SEQ ID NO. 1, 3 또는 5 중 1개와 적어도 80%, 90%, 95% 또는 99%의 서열 동일성을 가지거나 혹은 SEQ ID NO. 1, 3 또는 5 중 1개와 100%의 서열 동일성을 가지는 것이 바람직하다. 대체의 실시형태에 있어서, 핵산분자는, 엄격한 조건하에 있어서, SEQ ID NO. 1, 3 또는 5에 규정된 서열에 대하여 상보적인 서열을 가지는 폴리뉴클레오타이드에 혼성화하고, 케토오스-3-에피메라아제 활성을 가지는 단백질을 인코드하는 폴리뉴클레오타이드 서열을 포함한다. 몇 가지의 실시형태에 있어서, 효소의 천연 유래의 서열인, SEQ ID NO. 9, 10 또는 11의 서열을 가지는 폴리뉴클레오타이드를 구비한 핵산분자를 제공한다.
몇 가지의 실시형태에 있어서, 핵산분자는, 플라스미드 등의 벡터의 일부를 형성한다. 상기 서술의 핵산 서열에 더하여, 플라스미드는, 원핵성의 복제 기점(예를 들면, 대장균 OR1의 복제 기점), 자기 복제 서열, 동원체 서열, 프로모터 서열, 핵산 서열의 상류, 핵산 서열의 하류에 배치된 종료 서열, 항생물질 저항성 유전자, 및/또는, 분비 시그널 서열 등, 다른 요소를 포함한다. 자기 복제 서열을 포함한 벡터는 또, 효모 인공 염색체이다.
몇 가지의 대체의 실시형태에 있어서, 벡터는, 박테리오파지 등의 바이러스이며, 본 발명에 관한 핵산 서열에 더하여, 구조 단백질, 프로모터, 전사 활성화 인자 등, 박테리오파지의 복제를 행하는 핵산 서열을 포함한다.
본 발명에 관한 핵산분자는, 본 발명의 단백질을 합성하기 위하여, 숙주세포의 트랜스펙션 또는 형질전환을 위하여 사용되어도 된다. 적합한 숙주세포에는, 대장균 등의 원핵성 세포, 효모 세포 등의 진핵성 세포, 혹은 포유동물 또는 식물의 세포주가 포함된다. 숙주세포는, 일렉트로포레이션, 인산칼슘법, 유전자총기술(biolistic technique) 또는 바이러스벡터를 사용함으로써, 트랜스펙션 또는 형질전환을 행한다.
트랜스펙션 후, 본 발명의 핵산분자는, 필요에 따라서 전사되어, 번역된다. 몇 가지의 실시형태에 있어서, 합성 단백질이 숙주세포 내에 남겨져, 재조합 숙주세포의 배양물이 계속해서 사용된다. 다른 실시형태에 있어서, 예를 들면 벡터 내에 분비 신호가 존재하는 것에 의한 세포로부터의 분비나 혹은 숙주세포의 용해와 그로부터의 단백질의 정제에 의하여, 합성 단백질이 숙주세포로부터 추출된다.
본 발명에 관한 단백질은, 과당으로부터 알룰로오스로의 전환을 촉매하는 데에 사용된다. 몇 가지의 실시형태에 있어서, 단백질은 숙주세포 내에 존재하며, 전환 혼합물을 형성하기 때문에, 예를 들면 25℃~75℃의 온도에서 pH4~10에 있어서의 배양 등, 적합한 조건하에서 1~1000g/L의 농도로, 붕산염 버퍼과당 등의 과당 기질과 혼합한다. 이 전환 혼합물은 또한, 용매와, 임의로 예를 들면 에탄올, 톨루엔, 및 메탄올 등의 추가의 공용매(물 이외에)를 포함해도 된다. 과당 기질은 또한, 글루코오스 또는 수크로오스 등, 다른 당류를 포함해도 된다. 단백질은, 과당 기질의 알룰로오스로의 전환을 촉매한다. 실제로, 전환 혼합물 중의 모든 과당이 알룰로오스로 전환되는 것은 아니고, 통상, 계속해서 증발 및 결정화를 통하여 알룰로오스를 추출 및 정제하는 단계를 가진다. 혼합물 중의 나머지 과당은, 효모의 발효에 의하여 제거해도 된다.
대체의 실시형태에 있어서, 본 발명에 관한 단백질은, 정제된 형태로 제공되고, 또 완전하게 인비트로의 전환을 행하기 위하여 적합한 용매와 함께 과당 기질과 혼합된다. 일 실시형태에 있어서, 조건으로서, pH4~10, 30℃~70℃의 온도 범위, 10~95%w/v의 과당 농도, 및 용매로서의 물의 사용이 설정된다. 과당의 대체의 농도 범위로서, 20~95%, 30~95%, 40~95%, 50~95%, 60~95%, 70~95%, 75~95%가 포함되지만, 이에 한정되는 것은 아니다. 과당 기질은, 70~95%의 농도 범위에서 제공되는 것이 특히 바람직하다. 다른 적합한 실시형태에 있어서, 과당 농도는 75~95%이다.
케토오스의 전환 반응은 통상, 1~60%(w/v), 바람직하게는 약 5~50%의 기판농도를 이용하여 실시된다. 과당의 농도가 종래보다 높은 점을 제외하고 통상의 동작 조건하에서의 케토오스 전환 반응에 단백질을 사용할 수 있다는 점이, 본 발명의 특히 유리한 점이다. 따라서 본 발명에 관한 단백질에 의하여, 용적 생산성을 증가시킬 수 있다.
몇 가지의 실시형태에 있어서, 본 발명에 관한 단백질은, 고체기질에 고정화된다. 이로써, 효소의 사용 수명이 길어지고, 보다 소형의 고정상 반응기 내에 충전할 수 있게 되어, 오염이나 프로세스 조건의 변동에 대한 내성이 높아진다는 이점을 제공한다. 일례로서의 고체기질에는, 이온교환 수지 및 폴리머 봉지가 포함된다. 몇 가지의 실시형태에 있어서, 본 발명에 관한 단백질은, 듀오라이트 A568 수지에 고정화된다. 몇 가지의 실시형태에 있어서, 본 발명에 관한 단백질은, 약염기성 이온교환(즉, 단백질의 전하 및 수지 등의 기판의 전하에 근거하는 정전 상호작용)에 의하여, 기판 상에 고정화된다. 다른 실시형태에 있어서, 단백질은, 수지 등의 기판의 다공질 영역에 대한 비특이적 결합에 의하여 고정화된다.
다른 실시형태에 있어서, 본 발명은 알룰로오스의 생성방법에 관한 것이다. 이 방법에는, 이하의 단계가 포함된다.
1) SEQ ID NO: 1, SEQ ID NO: 3 또는 SEQ ID NO: 5와 적어도 70%의 서열 동일성을 가지는 핵산분자를 포함한 벡터를 제공하는 단계.
2) 상기 벡터로 컴피턴트 숙주세포를 형질전환하는 단계
3) 형질전환된 숙주세포를 임의로 배양하는 단계.
4) 형질전환된 세포를 과당 기질과 혼합하고, 과당으로부터 알룰로오스로의 전환을 허용하는 조건하에 유지하는 단계.
5) 증발 및 결정화 등, 종래 기술의 표준의 방법을 이용하여, 생성된 알룰로오스를 정제하는 단계.
대체의 실시형태에 있어서, 단계 4)는 생략된다. 혹은, 핵산분자에 의하여 인코드된 단백질을 형질전환된 숙주세포로부터 단리하여, 임의로 기판 상에 고정화한다. 그리고 이 단백질을 과당 기질과 혼합하여, 과당으로부터 알룰로오스로의 전환을 허용하는 조건하에 유지한다. 그 후, 단계 5)를 실시한다. 다른 실시형태에 있어서, 단계 2)를 생략하고, 대신에 단백질을 인비트로 번역으로 합성한다. 계속해서, 단백질을 단리하여, 과당 기질과 혼합한다.
본 발명에 관한 방법에 의하여 생성된 알룰로오스를, 인간 및/또는 동물의 소비에 제공되는 제품에 사용해도 된다. 몇 가지의 실시형태에 있어서, 이 제품은, 식품, 음료, 의약품, 영양제품, 스포츠제품 또는 화장품이어도 된다. 예를 들면, 이 제품이 식품인 경우, 이 식품은, 제과, 디저트제품, 시리얼제품, 구운 과자, 냉동 유제품, 육류, 유제품, 조미료, 스낵바, 스프, 드레싱, 혼합사료, 가공식품, 이유식, 다이어트식품, 시럽, 식품코팅, 건과일, 소스, 고기소스, 및 잼이나 젤리로 이루어지는 군으로부터 선택할 수 있다. 몇 가지의 실시형태에 있어서, 식품은, 제품의 표면에 형성되는 코팅 및 프로스팅으로서, 본 발명에 관한 방법에 의하여 생성된 알룰로오스를 포함해도 된다.
혹은, 제품이 음료인 경우, 이 음료는, 탄산음료, 비탄산음료, 과일맛음료, 과일주스, 차, 우유, 커피 등으로 이루어지는 군으로부터 선택할 수 있다.
실시예
실시예 1
본 예에서는, 3개의 추정 케토오스-3-에피메라아제 효소 중 1개를 발현하도록 숙주세포를 형질전환했다. 과당 기질에서 배양함으로써, 형질전환한 숙주세포의 케토오스-3-에피메라아제에 대하여 시험했다.
재료
붕산염 버퍼, 1M, pH8
i) 62g의 붕산을 1L의 탈이온수에 용해했다.
ii) 10M의 NaOH에 의하여 pH8로 조정했다.
iii) 1L 보틀에 넣어 4℃의 냉장고에 보존했다.
붕산염 버퍼 과당 기질
i) 970g의 액상 과당(77% DS)를 50ml의 붕산염 버퍼 pH8에 넣었다.
ii) 최종 용량이 1L가 되도록 물을 더했다.
iii) 5M의 NaOH에 의하여 pH8로 조정했다.
발현 배지 LB-4×2.8L 배플 장착 쉐이크 플라스크
i) 10g의 트립톤, 7g의 NaCl, 및 10g의 효모를 1L의 탈이온수에 넣었다.
ii) 오토 클레이브
방법
Genscript USA, Inc.에 의하여 합성 구축하기 위하여, 3개의 추정 케토오스-3-에피메라아제 유전자 서열 및 1개의 대조군 서열을 선택했다. 추정 케토오스-3-에피메라아제 서열을 인코드했다.
i) 클로스트리듐신덴스 ATCC 35704로부터의 가설 단백질 CLOSCI_02526(등록번호 ZP_02432281)(SEQ ID NO. 2)
ii) 클로스트리듐하이레몬아에 DSM 15053으로부터의 가설 단백질 CLOHYLEM_05645(등록번호 ZP_03778576.1)(SEQ ID NO. 4)
iii) 데스모스포라 8437종으로부터의 D-타가토오스 3-에피메라아제(등록번호 ZP_08466075)(SEQ ID NO. 6)
대조군 서열은, 클로스트리듐셀룰롤리티쿰 H10으로부터의 자일로오스아이소메라아제 단백질을 인코드했다(등록번호 YP_002505284)(SEQ ID NO. 8).
유전자는, 대장균(도 7~도 10 참조)에 있어서의 발현에 대하여 최적화된 서열을 가지도록 합성 구축하고, 결과적으로 얻어진 4개의 유전자를 각각, 발현 벡터 pET15b에 이식했다. 당업자에게 기지인 미생물 및 발현 벡터의 다른 조합으로도, 동일하게 실시할 수 있을 것으로 예측된다.
형질전환에 사용된 컴피턴트 세포는, 3ml의 LB배지(LB)에 대장균 BL21(DE3)를 식균하고, 37℃에서 오버나이트 박테리아를 번식시킴으로써 조제했다. 300ml의 LB에 이 3ml의 배양물을 식균하여, 0.7~1.0OD(600)로 쉐이킹하고, 37℃에서 세포를 성장시켰다. 광학밀도(OD)는, 통상의 분광 광도계의 600nm 파장에 있어서 1cm의 세포 내에서 측정했다. 세포는, 빙상에서 10분간 냉각한 후, 7500xg, 4℃에서 15분간, 원심했다. 배지를 제거하고, 세포를 300ml의 냉수에 재현탁했다. 원심을 반복하여, 세포를 150ml의 냉수에 재현탁했다. 원심을 다시 반복하여, 세포를 냉각·멸균한 약 2ml의 10% 글라이세롤에 현탁했다. 세포를 전회와 동일하게 원심하여, 냉각·멸균한 약 2ml의 10% 글라이세롤에 현탁했다. 현탁액은, 멸균한 에펜도르프관에 100μl 나누어, -80℃에서 보관했다.
계속해서 Genscript사로부터 제공되는 발현 벡터를 사용하여, 일렉트로포레이션으로 컴피턴트 대장균 BL21(DE3)을 형질전환하고, 암피실린 함유 LB 한천배지 상에서 양성 형질전환 세포를 선택했다. 1L의 LB를 4개의 2.8L 배플 장착 플라스크의 각각에 따르고, 오토 클레이브했다. 냉각한 후, 1ml의 100mg/l 암피실린을 무균으로 각 플라스크에 첨가하여, 상기 서술한 바와 같이 조제한 컴피턴트 세포의 오버나이트 배양물 2~3ml를 각 플라스크에 식균했다(발현 시스템마다 1개의 플라스크). 0.8-1.5의 OD를 달성하기 위하여, 세포를 200rpm의 쉐이킹에 의하여, 37℃에서 3시간 성장시켰다. 각 플라스크에는, 새롭게 조제한 1M의 아이소프로필 β-D-1-싸이오갈락토피라노사이드 용액을 1ml 첨가하고, 온도를 실온(즉 25-30℃)까지 내려, 약 5시간동안, 유도를 진행시켰다. 약 5000xg, 4℃에서 30분간, 세포를 원침하고, 상청액을 옮겼다. 세포 펠렛을 계량한 50ml의 원심관으로 옮겨, 세포량을 기록했다. 세포를 멸균한 글라이세롤(10%w/w) 수 ml 내에서 재현탁하고, -80℃에서 동결했다.
전체 세포를 붕산염 버퍼과당 기질에 혼합하고, Ca2+ 컬럼에 의한 DP 1-4법을 사용하여 HPCL에 의하여 해석을 행함으로써, 세포의 전환 활성을 확인했다. 250ml의 붕산염 버퍼과당 기질을 넣은 4개의 플라스크를 55℃로 승온하고, 동결 세포를 실온에 용해했다. 6500xg에서 세포를 작은 입자 형상으로 하고, 탈이온수 중에 재현탁했다. 2g(습중량)의 세포를 붕산염 버퍼과당 기질 중에 혼합하고, 1L의 배플 장착 플라스크 내에 있어서, 55℃에서 90rpm의 혼합에 의하여 배양했다. 0시간, 1시간, 2시간, 및 5시간동안 샘플을 취출하여, HPLC 해석에 적용했다.
HPLC 해석은, 0.1%(W/V)로 해석되는 20μL의 샘플을, 80℃로 유지된 Ca2+의 형태로, 0.1~1.5mL/분의 유량의 수계 이동상과 1~10μm의 입경 범위의 수지로 이루어지는 고정상으로 이루어지는 크로마토그래피 시스템에 주입하는 것을 포함한다. 굴절률 검출기에 의하여 피크를 검출 및 정량화하고, 기지의 기준에 의한 유지 시간에 근거하여 정성적으로 피크를 할당했다.
결과 및 검토
3개의 단백질 서열을, 케토오스-3-에피메라아제 단백질로서 시험하기 위하여 동정했다. 이들 단백질의 서열을 도 1~도 3(SEQ ID NO. 2, 4, 및 6)에 나타낸다. 대조군으로서 이용한 클로스트리듐셀룰롤리티쿰 H10으로부터의 자일로오스아이소메라아제는, 종래, 과당으로부터 알룰로오스를 생성하는 것으로서 제안되고 있었다. 이 아미노산 서열을 도 4에 나타낸다(SEQ ID NO. 8).
이들 단백질의 아미노산 서열을, 다른 기지의 케토오스-3-에피메라아제의 아미노산 서열과 정렬시키고, 이 정렬시킨 서열을 도 5에 나타낸다. 완전 보존 잔기는 하이라이트로 나타낸다. 이들의 서열 간에는 보존 잔기가 거의 존재하지 않으며, 1개의 서열로부터 다음 서열로 보존되는 잔기는 65% 미만이다.
등록번호 NP_535228 및 BAA24429를 각각 가지는 SEQ ID NO. 2, 4, 및 6과 SEQ ID NO. 8의 서열 간에서의 서열 동일성을 판정했다. 결과를 표 2에 나타낸다. 선택된 단백질 서열의 각각에 대하여, 기지의 케토오스-3-에피메라아제에는 40~63%의 서열 동일성이 인정되었다. 전체 서열의 서열 얼라인먼트에 근거하는 전체적인 강한 상동성은 인정되지 않았다. 선택된 단백질은, 합성 구축되어 Genscript사에 의한 시판의 발현 벡터 pEt15b에 이식된 대장균에 의하여, 발현을 위하여 최적화된 유전자를 가졌다. 각 구축물에 대하여 대장균 BL21(DE3)의 형질전환이 성공하여, 동결 보존한 각 균주를 발현 벡터와 함께 보존했다. 1L 스케일로 단백질 발현을 실시하고, 전체 세포를 조성 촉매로서 회수했다. 전환 활성을 확인하여, 도 6에 4개의 상이한 시험 균주에 의한 실험 중에 생성된 % DSB 알룰로오스를 나타낸다.
| % 동일 단백질 서열의 백분율 | ||||
| 유기체 | SEQ ID NO. |
아그로박테리움투메파시엔스 (등록번호 NP_535228) |
슈도모나스치코리이 (등록번호 BAA24429) |
클로스트리듐 셀룰롤리티쿰 (등록번호 NC_011898) |
| 클로스트리듐 신덴스 |
2 | 59 | 43 | 43 |
| 클로스트리듐 하이레몬아에 |
4 | 60 | 41 | 63 |
| 데스모스포라종 | 6 | 50 | 43 | 51 |
3개의 추정 케토오스-3-에피메라아제의 발현이 성공하고, 3개 모두 과당으로부터 알룰로오스로의 전환에 성공하여, 각 단백질이 실제로 케토오스-3-에피메라아제인 것을 확인했다.가장 활성인 단백질은 DS P3E(데스모스포라 8437종으로부터의 D-타가토오스 3-에피메라아제, SEQ ID NO. 6)이며, 용적 생산성 112g/L/시간에 대하여, 리터 당 8g의 세포습중량을 이용하여, 단 2시간에 750g/L의 과당 용액의 30%를 전환할 수 있었다.
실시예 2
본 예에서는, 스케일러빌리티를 판정하고, 추가적인 감각 조사 및 임상 조사를 행할 수 있도록 알룰로오스를 생성하기 위하여, 18L 스케일에 있어서 세포 성장 및 전환에 관하여 현재의 최고 조건을 실현했다. 전환에 이어서, 과당을 제거하는 최초의 세정 단계를 실시했다. 본 예는, 이 프로세스의 스케일러빌리티와, 스케일업에 따른 예기치 않은 문제와, 연구실에서 생성할 수 있는 알룰로오스의 타당한 양을 동정하는 것을 목적으로 하는 것이었다.
재료
아이소프로필싸이오갈락토피라노사이드(IPTG)
여과 멸균한 암피실린 수용액 100mg/ml
결정 과당 그린
액상 과당(77% DS)
성장 배지
i) 25g의 NaCl, 25g의 Staleydex 333(등록상표), 6g의 글라이세롤, 50g의 트립톤(Difco), 60g의 효모 추출물(Difco), 8g의 제1 인산칼륨, 8g의 제2 인산칼륨을 6l의 탈이온수에 넣었다
ii) 트리스 염기(고체)에 의해 pH7.8로 조정했다.
iii) 포일로 정상부를 덮은 6×2.8L 배플 장착 플라스크에서, 플라스크 당 1L의 오토 클레이브를 행했다.
트리스버퍼, 1M, pH8
iv) 121g을 1L의 탈이온수에 넣었다.
iv) HCl로 pH8로 조정했다.
iv) 1L 보틀에 넣어 4℃의 냉장고에서 보존했다.
방법
세포를 번식시키기 위하여, 100μg/ml의 암피실린을 보충한 5ml의 LB 배지의 6개의 오버나이트 배양을 개시했다. 이들 배양물에, 대장균 생성 균주(SEQ ID NO. 6의 단백질을 발현하는 BL21-DE3 pET15b-DS-P3E)를 식균하고, 37℃에서 오버나이트(~16시간) 성장시켰다. 6L의 성장 배지를 조제하여, 상기 서술한 바와 같이 오토 클레이브하고, 5ml의 오버나이트 배양물을 각 플라스크에 첨가하여, 190rpm, 37℃에서 4시간 이것을 쉐이킹했다. 새롭게 1M의 용액을 조제하여, 플라스크에 리터 당 1ml 첨가함으로써, 1mM의 IPTG를 각 플라스크에 첨가했다. 계속해서 14~16시간 쉐이킹을 행함으로써, 온도를 25℃까지 내렸다.
세포를 수집하기 위하여, Mod 322의 플로어 원심분리와 1L 보틀(800ml 이하의 배지를 충전)을 사용하여, 배양물을 6000rpm에서 20분간, 원심분리했다. 1질량%의 표백제를 첨가한 킬버킷 내에 이 배지를 옮겨, 30분간 두었다. 원심관을 계량하여, 세포 1g 당 3ml의 탈이온수를 관에 첨가했다. 균일한 세포 슬러리가 얻어질 때까지, 스패츌라 및 보텍스 지니를 사용하여 세포를 재현탁했다. 현탁액을 40ml의 원심관으로 옮겨, 6500xg에서 다시 작은 입자 형상으로 했다. 세정액을 킬버킷 내로 옮겨, 세포를 동일한 용량의 물로 재현탁했다.
제2 세포배치에 의하여, 세포의 증식 및 수집을 반복했다.
결정 과당 그린의 5갤런(18.9L) 버킷을 실온까지 승온하고, 16,506g의 결정 과당 그린을 18L의 교정눈금을 적용한 5갤런(18.9L)의 소독 완료된 플라스틱버킷에 첨가함으로써, 결정 과당 그린 전환기판을 조제했다. 상기 서술한 바와 같이 조제한 900ml의 1M 트리스 pH8.0을 버킷에 첨가하여, 18L 교정눈금까지 물을 넣고, 균일해질 때까지 오버헤드믹서를 사용하여 혼합했다. 이 혼합물과 미사용의 결정 과당 그린을 냉장실에 다시 넣어 보존했다.
18L의 교정눈금을 적용한 5갤런(18.9L)의 소독 완료된 플라스틱버킷 내에서, 17,460g의 액상 과당(77% DS) 및 500ml의 1M 트리스 pH8.0(상기 서술한 바와 같이 조제한 것)을 조합하여, 액상 과당 전환기판을 조제했다. 18L의 교정눈금까지 물을 넣고, 오버헤드믹서를 사용하여 균일해질 때까지 혼합했다.
전체 세포를 전환하기 위하여, 18L의 조제가 끝난 결정 과당 그린 전환기판을 수욕 중에서 55℃까지 승온하고, 오버헤드믹서에 의하여 약 150rpm로 부드럽게 혼합했다. 세포 수집으로 얻어진 재현탁 세포 페이스트를, 합계 100g의 습중량의 세포에 첨가했다. 5시간 후, 샘플을 취출하여, HPLC 해석을 행했다. 버킷 전체를 4℃로 냉장함으로써, 반응을 정지시켰다. 샘플에 대하여, 대장균, 대장균군, 및 TPC의 미생물 분석을 행했다.
전체 세포의 전환 프로세스는, 18L의 조제 액상 과당 전환기판에서 반복했다. 120g의 습중량의 세포를 사용하여, HPLC 해석을 위하여, 2시간 및 4시간동안 샘플을 추출했다.
효모발효를 사용하여, 결정 과당 그린 전환기판으로부터 과당을 제거했다. 결정 과당 그린 전환생성물은, 54L의 총 질량으로 결합된 알룰로오스 및 과당이 250g/L까지의 최종 농도가 되도록, 2배용의 물로 희석했다. 54L의 희석 혼합물을, 소독 완료된 4개의 5갤런(18.9L) 버킷에 나누고, 각 버킷이 약 13L가 되도록 했다. 버킷 중 2개는, 냉장고에 보존했다. 나머지 2개의 버킷은, 오버헤드믹서에서의 격렬한 교반과, 약 0.3VVM의 공기류를 보내기 위한 디퓨저를 구비한 9L/분의 공기펌프로부터의 통기로 조정했다. 120g의 건조 활성 빵효모(Fleishman 브랜드)를 각 버킷에 첨가하여, 혼합하고, DP1-4 알룰로오스 해석을 위하여 부정기 샘플링으로 2일간(~36시간) 통기했다. 버킷은 효모를 침전시키기 위하여, 오버나이트, 저온실로 옮겨졌다. 그 후, 상청액을 2개의 새로운 소독 완료된 청결한 버킷에 옮기고, 나머지 효모 부분을, 상기 서술한 바와 같이 조제한 2개의 냉장 결정 과당 그린 함유 버킷에 옮겼다. 교반 및 혼합 프로세스를 반복하고, 그 후, 효모를 제거했다. 효모발효 단계에 이어서, 약 45L의 상청액을 얻고, 3개의 소독 완료된 청결한 버킷에 무균 여과하고, 추가 처리를 실시하기 위하여 4℃에서 보존했다.
결과 및 검토
약 220g의 BL21(DE3) pET-15b-DS P3E 세포를 12L의 배양물로부터 얻어, 상기 서술한 바와 같이 2개의 18L의 생물 변환으로 나누었다. 따라서 전체 세포 생체촉매의 총 농도는, 결정 과당 그린 전환에 대해서는 5.6g/L이며, 액상 과당 전환에 대해서는 6.7g/L였다.
도 11은, 알룰로오스+과당의 백분율로서의 알룰로오스로서 계산하여, 쌍방의 전환이 급속하게 25%까지 달한 것을 나타내고 있다. 이는, 종래, 이 세포 종별과 동일한 스케일로 유사한 전환 배지를 사용하여 달성한 전환레벨보다 약간 낮지만, 매우 근접해 있다. 전환은, 액상 과당 기질에 의하여, 불과 2시간 후에는 이미 22%에 이르렀다.
각 18L 전환에 있어서, 약 3.3kg의 알룰로오스가 생성되었다. 2개의 기판의 사이에는 현저한 차이가 나타나지 않았다.
250ml의 스케일업에서는, 예기치 않았던 문제는 발생하지 않고, 예상했던 대로 진행했다.
미생물 시험은, 결과적으로, 살아있는 대장균은 존재하지 않아, 음성의 결과가 되었고, g당 3개 미만의 대장균군이 발견되었으며, 플레이트 전체 2개가 카운트되었다. 따라서 55℃의 온도와 DS시럽의 높은 백분율을 조합함으로써, 전체 세포의 생체촉매를 사멸시키는 데 충분했다.
새롭게 동정한 효소 DS P3E를 사용하여 과당으로부터 알룰로오스로의 생물 변환은 18L까지의 스케일업에 성공했다.
실시예 3
글루코오스 효모 추출 배지를 이용하는 pH제어 유가배양법을 사용하여, 발효 연구실에 있어서 2개의 10L 발효에 의하여, 새롭게 동정한 DS P3E 단백질(SEQ ID NO: 6)을 함유하는 대장균주를 생성했다. 이들의 발효는, 예상한 대로 진행되었다.
발효 배치 성장 및 유가배양동안, 세포는, 약 1시간의 배가 시간동안, 비약적으로 성장했다. 글루코오스 농도는, 약 5.5시간동안 약 9g/L에서 <1g/L로 떨어졌다(OD~28). 효소 생성의 유발위상에서는, OD가 계속하여 약 130까지 상승하고, 그 후에는 현저한 변화가 관찰되지 않았다. 원심분리에 의한 발효물의 수집에서는, 4.5kg(10lbs)의 습세포 페이스트 또는 약 1.1kg(2.5lbs)의 건조세포 중량이 얻어졌다.
과당 기질(836kg DS(건조고체) 베이스)을, RO수로 69% DS(920g/L)에 희석하고, 52℃까지 가열하여, pH를 7.8로 조정했다. 저레벨의 교반(~50rpm)을 이용하여, 반응 전체를 통하여 혼합을 촉진하고, 상기 서술한 바와 같이 발현한 4.5kg(습페이스트)의 전체 세포의 배치 전체를 반응에 첨가하여, 시간 0의 샘플을 채집했다. 이는, 전회 시험한 연구실 스케일 전환과 유사한 0.48g/L의 생체촉매 부하를 제공했지만, 920g/L로 기판농도는 보다 높아졌다. 4시간 및 16시간동안 샘플을 채집하여, HPLC로 해석했다.
DC의 상실은 관찰되지 않고, 반응하는 동안, 생물학적 생성물은 생성되지 않았다. 이 반응은, 16시간동안의 반응 종료 시, 30%까지의 알룰로오스의 평형치 부근까지 진행되었다. 4시간동안, 반응은 18%의 전환까지 이미 진행되고 있었다. 750g/L의 기판(실시예 1 및 2)과 함께 0.5g/L의 생체촉매를 사용하여 전회 얻어진 용적 전환율은, 46g/L*hr 또는, 단위 생체촉매 당 92g/L*hr/생체촉매 g이었다. 여기에서, 보다 높은 기판농도와 약간 낮은 온도(52℃ 대 55℃)를 사용하면, 용적 전환율은, 41g/L*hr 또는, 85g/L*hr/생체촉매의 g(4시간의 데이터 포인트를 사용하여 산출)이었다. 이로써, 에피메라아제 반응의 현저한 유연성을 실증했다. 반응이 16시간 후에 종료했을 때, 230kg의 알룰로오스가 알룰로오스:과당이 28:72인 혼합물 중에 존재했다.
실시예 4
4개의 상이한 효소(SEQ ID NO. 2, 4, 6, 및 8)에 의한 과당의 전환을, 750g/L에서 트리스버퍼과당 기질 상에서 비교했다. 25~30℃가 아니라, 16℃에서 세포를 유발하고, 전환율은 전회의 실험보다 낮았다. 200ml의 기판에 대하여, 500ml의 배플 장착 플라스크 내에서 2g의 습중량의 재현탁 세포를 첨가하여, 90rpm의 쉐이킹으로 55℃에서 배양했다. HPLC 해석을 행하기 위하여, 2시간 및 3.5시간동안 샘플을 채집했다. 결과를 도 12에 나타내는 데, 여기에서는 CC P3E는 SEQ ID NO. 8에 대응하고, CH P3E, CS P3E, 및 DS P3E가 각각, SEQ ID NO. 4, 2, 및 6에 대응한다. 이 실험에 있어서, SEQ ID NO. 2, 4, 6 또는 8에 규정의 단백질의 하나를 발현하는 4개의 균주는 모두, 3.5시간 후에 기판의 약 5%를 알룰로오스로 전환하는 대략 동일한 활성 레벨을 가지고 나타났다.
실시예 5
본 예에서는, 고정화 효소를 사용한 알룰로오스 생성의 제1 시도를 행했다. 본 예는, 효소의 이용을 개선하는 것을 목적으로 하고 있다.
재료
Codexis로부터 조제된 동결 건조 효소 분말, Lot D13007 또는 D13008
데스모스포라종, 사이코스-3-에피메라아제
듀오라이트 A568(Dow)
앰버라이트 XAD2(Sigma)
트리스버퍼 1M
i) 1M의 농도로 용해함으로써 수 중에 조제
ii) HCl로 pH를 8.0로 조정
iii) 사용 전에 100mM에 희석
결정 과당 그린, 80%의 건조고체에서 이하의 조성을 가진다.
i) 90% DSB 과당
ii) 7% DSB 덱스트로오스
iii) 3% DP2+
iv) 그 외의 단당류
MnCl2(Sigma)
방법
1) 소규모의 고정화
고정화의 효율을 시험하기 위하여, 약 30ml의 질량의 피복컬럼(11mm×300mm의 컬럼 치수) 내에서 작은 고정상 반응을 실시했다. XAD2 수지 및 A568 수지의 양방을 물로 수 회 세정함으로써, 미립자(즉, 수지 작성 시의 부산물인 수지 미립자(즉, 균열된/깨진 입자))와 작성 시의 잔류물을 제거했다. 2g의 동결건조 효소(즉, 에피메라아제)를 약 50ml의 물에 용해하고, 2개의 앨리컷에 나누었다. pH를 측정하여, 6.5라고 판정했다. 약 30ml의 각 수지를, 실온에서 약 1시간, 가벼운 교반에 의하여 1개의 앨리컷의 에피메라아제 용액에 의하여 배양했다. 그 후, 수지를 피복컬럼 내에 채우고, 연동펌프를 사용하여 2시간, 고정상을 통하여 에피메라아제 용액을 더욱 순환시켰다. 그 후, 10베드용적의 트리스버퍼 100mM, pH8.0으로 컬럼을 세정했다. 이 시점에서의 폐수는 투명하게 보이며, A280에서는 분광학적으로 측정된 단백질은 없었다. 결정 과당 그린을 RO수로 60% DS까지 희석하여, pH를 8.0까지 조정한 후, 28ppm의 MnCl2 및 10mM의 트리스버퍼 pH8.0을 첨가함으로써, 결정 과당 그린 공급물을 조제했다.
그 후, 재순환 수욕으로 피복컬럼을 57℃의 온도까지 가열함으로써, 1시간당 8베드용적(BV/h)의 비율로, 공급물을 30ml 고정상 반응기를 통하여 퍼 올렸다. 반응기의 폐액을 회수하고, FT-IR로 해석하여 알룰로오스와 과당의 상대농도를 구했다. 이 프로세스를 합계 5일간 계속했다. 이 공급률은, 전환율을 유지하기 위하여, 시험적 생성공정을 통하여 1시간당 8베드용적에서 1시간당 6베드용적으로 조정했다.
2) 300ml 고정상 반응기에 대한 스케일업
고정화의 스케일업된 효율을 시험하기 위하여, 약 300ml의 질량의 피복컬럼 내(25mm×600mm 컬럼 치수)에 의하여 대형의 고정상 반응기를 작성했다. XAD2 수지 및 A568 수지를 물로 수 회 세정함으로써, 미립자나 작성 시의 잔류물을 제거했다. 10g의 동결건조 에피메라아제를 약 100ml의 물에 용해했다. 약 300ml의 A568 수지를 300ml의 피복컬럼에 채우고, 연동펌프를 사용하여 실온에서 약 2시간, 고정상을 통하여 에피메라아제 용액을 순환시켰다. 그 후, 실온에서, 컬럼을 5베드용적의 트리스버퍼 100mM, pH8.0으로 세정했다. 이 시점에서의 폐수는 투명하게 보이며, A280에서는 분광학적으로 측정된 단백질은 없었다. 결정 과당 그린을 RO수로 60% DS까지 희석하고, pH를 8.0까지 조정한 후, 28ppm의 MnCl2 및 10mM의 트리스버퍼 pH8.0을 첨가함으로써, 결정 과당 그린 공급물을 조제했다. 그 후, 재순환 수욕으로 피복컬럼을 57℃의 온도까지 가열함으로써, 1시간당 8베드용적(BV/h)의 비율로, 공급물을 30ml 고정상 반응기를 통하여 퍼 올렸다. 반응기의 폐액을 회수하여, FT-IR로 해석하고 알룰로오스와 과당의 상대농도를 구했다. 이 프로세스를 합계 4일동안 계속했다. 이 공급률은, 시험적 생성공정을 통하여 1시간당 8베드용적으로부터 1시간당 2베드용적으로 조정했다. 또한, 컬럼 2를 2주간, 실온에 둔 후, 컬럼 보관 중인 에피메라아제 안정성의 판정을 재개했다.
결과 및 검토
XAD2를 넣은 30ml의 컬럼에서는, 어느 샘플에서도 현저한 전환이 발견되지 않았고, 따라서 새로운 해석을 실시하지 않았다. 그러나, Dowex의 A568에서 현저한 전환이 관찰되었다. 도 13은, A568 수지를 넣은 30ml의 고정상 반응기에 있어서의 반응의 시간 경과를 나타내고 있다. 불과 1g의 에피메라아제를 함유하는 고정상 전환을 120시간 계속함으로써, 4kg가 넘는 알룰로오스를 생성했다. 전환율은, 120시간을 넘기면 서서히 저하했으며, 이를 보상하기 위하여 컬럼의 유량을 72시간동안 저하시켰다. 반응 중, 알룰로오스의 평형 근방 농도가 얻어졌다.
도 14는, A568 수지에 의한 300ml의 반응공정을 나타내고 있다. 이용 가능한 공급물의 양(72시간에 86l의 공급물을 사용)에 한도가 있기 때문에, 유량은 24시간 후에는 저하되어 있었다. 72시간동안, 10g의 에피메라아제로부터 20kg 초과의 알룰로오스를 생성했다. 어떠한 성능 저하가 관찰되었지만, 72시간 종료 시에도 여전히 전환율은 높은 상태인 채였다.
용액 중의 에피메라아제의 안정성은, 플라스크 반응 이전에 판정되었다. 53℃에서 8시간 이내에 90%가 넘는 활성이 손상되었다. 본 예에 있어서, 전환은 57℃에서 실시되었다. 반응률, 평형비, 및 미생물 안정성의 관점에서, 온도는 보다 높은 편이 유리하다. 본 예에서는, 120시간 후에도 현저한 에피메라아제 활성이 유지되었다.
공급이 한정적이었던 대규모의 반응에 있어서, 10g의 에피메라아제를 사용함으로써 20kg의 알룰로오스를 생성했으며, 결과적으로 순에피메라아제의 투여량은 0.05%(m/m)였다. 소규모의 반응에서는, 1g의 에피메라아제로부터 4.8kg의 알룰로오스가 생성되어, 결과적으로 순에피메라아제의 투여량은 0.02%(m/m)였다. 표준적인 과당의 생성에 있어서, 고정화된 글루코아이소메라아제는 0.01~0.005%(m/)의 비율로 사용되지만, 이 생성은 6~12개월이 걸리는 작업이다.
SEQUENCE LISTING - Rule 13ter
<110> Tate & Lyle Ingredients Americas LLC
<120> A Protein
<130> PN805090WO
<140> PCT/GB2013/052531
<141> 2013-09-27
<150> US 61/706,338
<151> 2012-09-27
<150> GB 1220554.8
<151> 2012-11-15
<160> 15
<170> PatentIn version 3.5
<210> 1
<211> 903
<212> DNA
<213> Clostridium scindens
<220>
<221> CDS
<222> (4)..(888)
<400> 1
cat atg aat cgt att ggc att ttt atg aat ttt tgg gtg aag aac tgg 48
Met Asn Arg Ile Gly Ile Phe Met Asn Phe Trp Val Lys Asn Trp
1 5 10 15
gac gct gac cac gtt aag tac atc aag aag gtg tcg ggc ctg ggc ttt 96
Asp Ala Asp His Val Lys Tyr Ile Lys Lys Val Ser Gly Leu Gly Phe
20 25 30
gat att ctg gaa ttt cag gca caa gct ctg ctg gaa atg gat aaa tct 144
Asp Ile Leu Glu Phe Gln Ala Gln Ala Leu Leu Glu Met Asp Lys Ser
35 40 45
cgt atg gac gaa gtg cgc cag gcg gcc aag gat aac ggc att gaa ctg 192
Arg Met Asp Glu Val Arg Gln Ala Ala Lys Asp Asn Gly Ile Glu Leu
50 55 60
acc tat tct ctg ggt ctg aat ccg aaa tac gat gtg gca agt ccg gac 240
Thr Tyr Ser Leu Gly Leu Asn Pro Lys Tyr Asp Val Ala Ser Pro Asp
65 70 75
gct aag gtt cgt gaa ggc ggt atc gaa tat ctg aaa cgt att gtg gaa 288
Ala Lys Val Arg Glu Gly Gly Ile Glu Tyr Leu Lys Arg Ile Val Glu
80 85 90 95
cgc atc ggc tac atg gaa ggc aag ctg ctg tca ggc gtt aac tat gcg 336
Arg Ile Gly Tyr Met Glu Gly Lys Leu Leu Ser Gly Val Asn Tyr Ala
100 105 110
ggc tgg ggt tcg ccg gat tac att gtc gat gac aaa agc gaa att gtg 384
Gly Trp Gly Ser Pro Asp Tyr Ile Val Asp Asp Lys Ser Glu Ile Val
115 120 125
gaa cat agc atc gaa agc gtg cgt cag gtc atc aaa acc gcc gaa gat 432
Glu His Ser Ile Glu Ser Val Arg Gln Val Ile Lys Thr Ala Glu Asp
130 135 140
tat gac gtg acg tac tgc gtt gaa gtg gtt aac cgc ttt gaa ggc att 480
Tyr Asp Val Thr Tyr Cys Val Glu Val Val Asn Arg Phe Glu Gly Ile
145 150 155
gtt atg aat acc gcg aaa gaa gcc att gaa tat gtc aaa caa atc gat 528
Val Met Asn Thr Ala Lys Glu Ala Ile Glu Tyr Val Lys Gln Ile Asp
160 165 170 175
agc gac aag att ggt atc ctg ctg gat acg tac cac atg aac atc gaa 576
Ser Asp Lys Ile Gly Ile Leu Leu Asp Thr Tyr His Met Asn Ile Glu
180 185 190
gaa ggc agt att ggt gat gcg atc cgt tcc gtt ggc ggt tat ctg aaa 624
Glu Gly Ser Ile Gly Asp Ala Ile Arg Ser Val Gly Gly Tyr Leu Lys
195 200 205
aat ttc cac acg ggc gaa aac aat cgc gtc gtg ccg ggc aag ggt cat 672
Asn Phe His Thr Gly Glu Asn Asn Arg Val Val Pro Gly Lys Gly His
210 215 220
ctg gat tgg gac gaa att ttt ggc gca ctg cac gat att gac tac cag 720
Leu Asp Trp Asp Glu Ile Phe Gly Ala Leu His Asp Ile Asp Tyr Gln
225 230 235
ggt cgc atc gtc tcc gaa ccg ttc gtg caa atg ggc ggt gaa gtg gct 768
Gly Arg Ile Val Ser Glu Pro Phe Val Gln Met Gly Gly Glu Val Ala
240 245 250 255
cgt gat atc aaa gtt tgg cgc gat ctg gtc gaa gac ccg agc gaa gaa 816
Arg Asp Ile Lys Val Trp Arg Asp Leu Val Glu Asp Pro Ser Glu Glu
260 265 270
gtt ctg gat gaa gaa gcg cgt ttt ctg ctg aat ttc gaa aaa gac atg 864
Val Leu Asp Glu Glu Ala Arg Phe Leu Leu Asn Phe Glu Lys Asp Met
275 280 285
att cgc aag cac tat ggt atc gcc taaagatctg gatcc 903
Ile Arg Lys His Tyr Gly Ile Ala
290 295
<210> 2
<211> 295
<212> PRT
<213> Clostridium scindens
<400> 2
Met Asn Arg Ile Gly Ile Phe Met Asn Phe Trp Val Lys Asn Trp Asp
1 5 10 15
Ala Asp His Val Lys Tyr Ile Lys Lys Val Ser Gly Leu Gly Phe Asp
20 25 30
Ile Leu Glu Phe Gln Ala Gln Ala Leu Leu Glu Met Asp Lys Ser Arg
35 40 45
Met Asp Glu Val Arg Gln Ala Ala Lys Asp Asn Gly Ile Glu Leu Thr
50 55 60
Tyr Ser Leu Gly Leu Asn Pro Lys Tyr Asp Val Ala Ser Pro Asp Ala
65 70 75 80
Lys Val Arg Glu Gly Gly Ile Glu Tyr Leu Lys Arg Ile Val Glu Arg
85 90 95
Ile Gly Tyr Met Glu Gly Lys Leu Leu Ser Gly Val Asn Tyr Ala Gly
100 105 110
Trp Gly Ser Pro Asp Tyr Ile Val Asp Asp Lys Ser Glu Ile Val Glu
115 120 125
His Ser Ile Glu Ser Val Arg Gln Val Ile Lys Thr Ala Glu Asp Tyr
130 135 140
Asp Val Thr Tyr Cys Val Glu Val Val Asn Arg Phe Glu Gly Ile Val
145 150 155 160
Met Asn Thr Ala Lys Glu Ala Ile Glu Tyr Val Lys Gln Ile Asp Ser
165 170 175
Asp Lys Ile Gly Ile Leu Leu Asp Thr Tyr His Met Asn Ile Glu Glu
180 185 190
Gly Ser Ile Gly Asp Ala Ile Arg Ser Val Gly Gly Tyr Leu Lys Asn
195 200 205
Phe His Thr Gly Glu Asn Asn Arg Val Val Pro Gly Lys Gly His Leu
210 215 220
Asp Trp Asp Glu Ile Phe Gly Ala Leu His Asp Ile Asp Tyr Gln Gly
225 230 235 240
Arg Ile Val Ser Glu Pro Phe Val Gln Met Gly Gly Glu Val Ala Arg
245 250 255
Asp Ile Lys Val Trp Arg Asp Leu Val Glu Asp Pro Ser Glu Glu Val
260 265 270
Leu Asp Glu Glu Ala Arg Phe Leu Leu Asn Phe Glu Lys Asp Met Ile
275 280 285
Arg Lys His Tyr Gly Ile Ala
290 295
<210> 3
<211> 885
<212> DNA
<213> Clostridium hylemonae
<220>
<221> CDS
<222> (4)..(870)
<400> 3
cat atg aaa cac ggt atc tat tac gcc tat tgg gaa caa gaa tgg gca 48
Met Lys His Gly Ile Tyr Tyr Ala Tyr Trp Glu Gln Glu Trp Ala
1 5 10 15
gca gac tac aaa cgc tat gtg gaa aaa gtg gca aaa ctg ggc ttc gat 96
Ala Asp Tyr Lys Arg Tyr Val Glu Lys Val Ala Lys Leu Gly Phe Asp
20 25 30
att ctg gaa atc ggc gcc ggt ccg ctg ccg gaa tat gca gaa cag gac 144
Ile Leu Glu Ile Gly Ala Gly Pro Leu Pro Glu Tyr Ala Glu Gln Asp
35 40 45
gtt aaa gaa ctg aaa aag tgc gct caa gat aac ggc att acc ctg acg 192
Val Lys Glu Leu Lys Lys Cys Ala Gln Asp Asn Gly Ile Thr Leu Thr
50 55 60
gcg ggc tac ggt ccg acc ttt aac cat aat atc ggc agc tct gat gct 240
Ala Gly Tyr Gly Pro Thr Phe Asn His Asn Ile Gly Ser Ser Asp Ala
65 70 75
ggt gtg cgt gaa gaa gcg ctg gaa tgg tat aaa cgc ctg ttc gaa gtt 288
Gly Val Arg Glu Glu Ala Leu Glu Trp Tyr Lys Arg Leu Phe Glu Val
80 85 90 95
ctg gcc gaa ctg gac att cac ctg atc ggc ggt gca ctg tat agt tac 336
Leu Ala Glu Leu Asp Ile His Leu Ile Gly Gly Ala Leu Tyr Ser Tyr
100 105 110
tgg ccg gtc gat ttt gct aac gcg gac aaa acg gaa gat tgg aag tgg 384
Trp Pro Val Asp Phe Ala Asn Ala Asp Lys Thr Glu Asp Trp Lys Trp
115 120 125
tcc gtg gag ggt atg cag cgt ctg gcc ccg gcg gcg gca aaa tac gat 432
Ser Val Glu Gly Met Gln Arg Leu Ala Pro Ala Ala Ala Lys Tyr Asp
130 135 140
att aac ctg ggt atg gaa gtt ctg aat cgc ttt gaa tca cat atc ctg 480
Ile Asn Leu Gly Met Glu Val Leu Asn Arg Phe Glu Ser His Ile Leu
145 150 155
aat acc gcc gaa gaa ggc gtc aaa ttc gtg gaa gaa gtt ggt atg gac 528
Asn Thr Ala Glu Glu Gly Val Lys Phe Val Glu Glu Val Gly Met Asp
160 165 170 175
aac gtg aag gtt atg ctg gat acg ttc cac atg aat att gaa gaa caa 576
Asn Val Lys Val Met Leu Asp Thr Phe His Met Asn Ile Glu Glu Gln
180 185 190
tcg att ggc ggt gcc atc cgt cgc gca ggc aaa ctg ctg ggt cat ttt 624
Ser Ile Gly Gly Ala Ile Arg Arg Ala Gly Lys Leu Leu Gly His Phe
195 200 205
cac acc ggc gaa tgt aat cgt atg gtg ccg ggc aag ggt cgt att ccg 672
His Thr Gly Glu Cys Asn Arg Met Val Pro Gly Lys Gly Arg Ile Pro
210 215 220
tgg cgc gaa atc ggt gac gct ctg cgt gat atc ggc tac gac ggt acg 720
Trp Arg Glu Ile Gly Asp Ala Leu Arg Asp Ile Gly Tyr Asp Gly Thr
225 230 235
gca gtc atg gaa ccg ttc gtg cgt atg ggt ggt cag gtt ggt gca gat 768
Ala Val Met Glu Pro Phe Val Arg Met Gly Gly Gln Val Gly Ala Asp
240 245 250 255
att aaa gtc tgg cgt gac atc tct cgc ggt gcc gat gaa gca cag ctg 816
Ile Lys Val Trp Arg Asp Ile Ser Arg Gly Ala Asp Glu Ala Gln Leu
260 265 270
gat gac gat gct cgt cgc gcg ctg gaa ttt caa cgc tat atg ctg gaa 864
Asp Asp Asp Ala Arg Arg Ala Leu Glu Phe Gln Arg Tyr Met Leu Glu
275 280 285
tgg aag taaagatctg gatcc 885
Trp Lys
<210> 4
<211> 289
<212> PRT
<213> Clostridium hylemonae
<400> 4
Met Lys His Gly Ile Tyr Tyr Ala Tyr Trp Glu Gln Glu Trp Ala Ala
1 5 10 15
Asp Tyr Lys Arg Tyr Val Glu Lys Val Ala Lys Leu Gly Phe Asp Ile
20 25 30
Leu Glu Ile Gly Ala Gly Pro Leu Pro Glu Tyr Ala Glu Gln Asp Val
35 40 45
Lys Glu Leu Lys Lys Cys Ala Gln Asp Asn Gly Ile Thr Leu Thr Ala
50 55 60
Gly Tyr Gly Pro Thr Phe Asn His Asn Ile Gly Ser Ser Asp Ala Gly
65 70 75 80
Val Arg Glu Glu Ala Leu Glu Trp Tyr Lys Arg Leu Phe Glu Val Leu
85 90 95
Ala Glu Leu Asp Ile His Leu Ile Gly Gly Ala Leu Tyr Ser Tyr Trp
100 105 110
Pro Val Asp Phe Ala Asn Ala Asp Lys Thr Glu Asp Trp Lys Trp Ser
115 120 125
Val Glu Gly Met Gln Arg Leu Ala Pro Ala Ala Ala Lys Tyr Asp Ile
130 135 140
Asn Leu Gly Met Glu Val Leu Asn Arg Phe Glu Ser His Ile Leu Asn
145 150 155 160
Thr Ala Glu Glu Gly Val Lys Phe Val Glu Glu Val Gly Met Asp Asn
165 170 175
Val Lys Val Met Leu Asp Thr Phe His Met Asn Ile Glu Glu Gln Ser
180 185 190
Ile Gly Gly Ala Ile Arg Arg Ala Gly Lys Leu Leu Gly His Phe His
195 200 205
Thr Gly Glu Cys Asn Arg Met Val Pro Gly Lys Gly Arg Ile Pro Trp
210 215 220
Arg Glu Ile Gly Asp Ala Leu Arg Asp Ile Gly Tyr Asp Gly Thr Ala
225 230 235 240
Val Met Glu Pro Phe Val Arg Met Gly Gly Gln Val Gly Ala Asp Ile
245 250 255
Lys Val Trp Arg Asp Ile Ser Arg Gly Ala Asp Glu Ala Gln Leu Asp
260 265 270
Asp Asp Ala Arg Arg Ala Leu Glu Phe Gln Arg Tyr Met Leu Glu Trp
275 280 285
Lys
<210> 5
<211> 885
<212> DNA
<213> Desmospora sp.8437
<220>
<221> CDS
<222> (4)..(870)
<400> 5
cat atg aaa tac ggt gtc tac ttt gct tac tgg gaa gat tcg tgg gat 48
Met Lys Tyr Gly Val Tyr Phe Ala Tyr Trp Glu Asp Ser Trp Asp
1 5 10 15
gtt gac ttt gaa aaa tac gtt cgc aag gtg aaa aaa ctg ggc ttt gat 96
Val Asp Phe Glu Lys Tyr Val Arg Lys Val Lys Lys Leu Gly Phe Asp
20 25 30
att ctg gaa gtt gca gca ctg ggt ctg gtc aac ctg ccg gaa gaa aaa 144
Ile Leu Glu Val Ala Ala Leu Gly Leu Val Asn Leu Pro Glu Glu Lys
35 40 45
ctg gaa cgt ctg aag cag ctg gcg gaa caa cat gac att atc ctg acc 192
Leu Glu Arg Leu Lys Gln Leu Ala Glu Gln His Asp Ile Ile Leu Thr
50 55 60
gcc ggc att ggt ctg ccg aaa gaa tat gat gtc agc tct acg gac aaa 240
Ala Gly Ile Gly Leu Pro Lys Glu Tyr Asp Val Ser Ser Thr Asp Lys
65 70 75
aaa gtg cgt cgc aat ggc atc tcc ttt atg aaa aag gtt atg gat gca 288
Lys Val Arg Arg Asn Gly Ile Ser Phe Met Lys Lys Val Met Asp Ala
80 85 90 95
atg cat cag gct ggt att cac cgt att ggc ggc acc gtg tat agc tac 336
Met His Gln Ala Gly Ile His Arg Ile Gly Gly Thr Val Tyr Ser Tyr
100 105 110
tgg ccg gtt gat tac agt tgc tcc ttc gac aaa ccg gcg gtt cgc aag 384
Trp Pro Val Asp Tyr Ser Cys Ser Phe Asp Lys Pro Ala Val Arg Lys
115 120 125
cac tca att gaa tcg gtc cgt gaa ctg gcg gaa tat gcc cgc cag tac 432
His Ser Ile Glu Ser Val Arg Glu Leu Ala Glu Tyr Ala Arg Gln Tyr
130 135 140
aac att acc ctg ctg atc gaa acg ctg aac cgc ttt gaa caa ttc ctg 480
Asn Ile Thr Leu Leu Ile Glu Thr Leu Asn Arg Phe Glu Gln Phe Leu
145 150 155
ctg aat gat gcc gaa gaa gcg gtt gcc tat gtc aaa gaa gtg gat gaa 528
Leu Asn Asp Ala Glu Glu Ala Val Ala Tyr Val Lys Glu Val Asp Glu
160 165 170 175
ccg aac gtc aag gtg atg ctg gac acc ttc cac atg aac atc gaa gaa 576
Pro Asn Val Lys Val Met Leu Asp Thr Phe His Met Asn Ile Glu Glu
180 185 190
gat cac atc gca gac gct atc cgt tac acg ggc gat cat ctg ggt cag 624
Asp His Ile Ala Asp Ala Ile Arg Tyr Thr Gly Asp His Leu Gly Gln
195 200 205
ctg cac atc ggc gaa gcc aac cgc aaa gtg ccg ggc aag ggt agt atg 672
Leu His Ile Gly Glu Ala Asn Arg Lys Val Pro Gly Lys Gly Ser Met
210 215 220
ccg tgg acc gaa att ggc caa gca ctg aaa gat atc cgt tat gac ggt 720
Pro Trp Thr Glu Ile Gly Gln Ala Leu Lys Asp Ile Arg Tyr Asp Gly
225 230 235
tac gtg gtt atg gaa ccg ttc att aaa acc ggc ggt cag gtt ggc cgt 768
Tyr Val Val Met Glu Pro Phe Ile Lys Thr Gly Gly Gln Val Gly Arg
240 245 250 255
gat atc aaa ctg tgg cgc gac ctg agc ggt aat gca acg gaa gaa caa 816
Asp Ile Lys Leu Trp Arg Asp Leu Ser Gly Asn Ala Thr Glu Glu Gln
260 265 270
ctg gat cgc gaa ctg gct gaa tct ctg gaa ttt gtg aaa gca gct ttc 864
Leu Asp Arg Glu Leu Ala Glu Ser Leu Glu Phe Val Lys Ala Ala Phe
275 280 285
ggt gaa taaagatctg gatcc 885
Gly Glu
<210> 6
<211> 289
<212> PRT
<213> Desmospora sp.8437
<400> 6
Met Lys Tyr Gly Val Tyr Phe Ala Tyr Trp Glu Asp Ser Trp Asp Val
1 5 10 15
Asp Phe Glu Lys Tyr Val Arg Lys Val Lys Lys Leu Gly Phe Asp Ile
20 25 30
Leu Glu Val Ala Ala Leu Gly Leu Val Asn Leu Pro Glu Glu Lys Leu
35 40 45
Glu Arg Leu Lys Gln Leu Ala Glu Gln His Asp Ile Ile Leu Thr Ala
50 55 60
Gly Ile Gly Leu Pro Lys Glu Tyr Asp Val Ser Ser Thr Asp Lys Lys
65 70 75 80
Val Arg Arg Asn Gly Ile Ser Phe Met Lys Lys Val Met Asp Ala Met
85 90 95
His Gln Ala Gly Ile His Arg Ile Gly Gly Thr Val Tyr Ser Tyr Trp
100 105 110
Pro Val Asp Tyr Ser Cys Ser Phe Asp Lys Pro Ala Val Arg Lys His
115 120 125
Ser Ile Glu Ser Val Arg Glu Leu Ala Glu Tyr Ala Arg Gln Tyr Asn
130 135 140
Ile Thr Leu Leu Ile Glu Thr Leu Asn Arg Phe Glu Gln Phe Leu Leu
145 150 155 160
Asn Asp Ala Glu Glu Ala Val Ala Tyr Val Lys Glu Val Asp Glu Pro
165 170 175
Asn Val Lys Val Met Leu Asp Thr Phe His Met Asn Ile Glu Glu Asp
180 185 190
His Ile Ala Asp Ala Ile Arg Tyr Thr Gly Asp His Leu Gly Gln Leu
195 200 205
His Ile Gly Glu Ala Asn Arg Lys Val Pro Gly Lys Gly Ser Met Pro
210 215 220
Trp Thr Glu Ile Gly Gln Ala Leu Lys Asp Ile Arg Tyr Asp Gly Tyr
225 230 235 240
Val Val Met Glu Pro Phe Ile Lys Thr Gly Gly Gln Val Gly Arg Asp
245 250 255
Ile Lys Leu Trp Arg Asp Leu Ser Gly Asn Ala Thr Glu Glu Gln Leu
260 265 270
Asp Arg Glu Leu Ala Glu Ser Leu Glu Phe Val Lys Ala Ala Phe Gly
275 280 285
Glu
<210> 7
<211> 897
<212> DNA
<213> Clostridium cellulolyticum
<220>
<221> CDS
<222> (4)..(882)
<400> 7
cat atg aag cac ggc atc tat tac gcc tat tgg gaa caa gaa tgg gaa 48
Met Lys His Gly Ile Tyr Tyr Ala Tyr Trp Glu Gln Glu Trp Glu
1 5 10 15
gca gac tac aag tat tac atc gaa aag gtt gcg aag ctg ggt ttt gat 96
Ala Asp Tyr Lys Tyr Tyr Ile Glu Lys Val Ala Lys Leu Gly Phe Asp
20 25 30
att ctg gaa atc gcg gcc tca ccg ctg ccg ttt tat tcg gac att cag 144
Ile Leu Glu Ile Ala Ala Ser Pro Leu Pro Phe Tyr Ser Asp Ile Gln
35 40 45
atc aat gaa ctg aaa gcg tgc gcg cat ggc aac ggt att acc ctg acg 192
Ile Asn Glu Leu Lys Ala Cys Ala His Gly Asn Gly Ile Thr Leu Thr
50 55 60
gtg ggc cac ggt ccg agc gcg gaa caa aat ctg agc agc ccg gac ccg 240
Val Gly His Gly Pro Ser Ala Glu Gln Asn Leu Ser Ser Pro Asp Pro
65 70 75
gac atc cgt aaa aac gca aag gct ttc tat acc gat ctg ctg aaa cgc 288
Asp Ile Arg Lys Asn Ala Lys Ala Phe Tyr Thr Asp Leu Leu Lys Arg
80 85 90 95
ctg tac aag ctg gac gtt cat ctg att ggc ggt gcc ctg tat tct tac 336
Leu Tyr Lys Leu Asp Val His Leu Ile Gly Gly Ala Leu Tyr Ser Tyr
100 105 110
tgg ccg atc gat tac acc aag acg atc gat aag aag ggc gac tgg gaa 384
Trp Pro Ile Asp Tyr Thr Lys Thr Ile Asp Lys Lys Gly Asp Trp Glu
115 120 125
cgt agt gtt gaa tcc gtc cgc gaa gtg gcc aag gtt gcg gaa gcc tgc 432
Arg Ser Val Glu Ser Val Arg Glu Val Ala Lys Val Ala Glu Ala Cys
130 135 140
ggt gtc gat ttt tgt ctg gaa gtg ctg aac cgt ttc gaa aat tac ctg 480
Gly Val Asp Phe Cys Leu Glu Val Leu Asn Arg Phe Glu Asn Tyr Leu
145 150 155
att aac acc gca cag gaa ggc gtc gat ttt gtg aaa caa gtt gac cat 528
Ile Asn Thr Ala Gln Glu Gly Val Asp Phe Val Lys Gln Val Asp His
160 165 170 175
aac aat gtc aag gtg atg ctg gat acg ttc cac atg aat atc gaa gaa 576
Asn Asn Val Lys Val Met Leu Asp Thr Phe His Met Asn Ile Glu Glu
180 185 190
gac agt att ggc ggt gcg atc cgt acc gcc ggc tcc tat ctg ggt cat 624
Asp Ser Ile Gly Gly Ala Ile Arg Thr Ala Gly Ser Tyr Leu Gly His
195 200 205
ctg cac acg ggc gaa tgc aat cgc aaa gtt ccg ggc cgt ggt cgc att 672
Leu His Thr Gly Glu Cys Asn Arg Lys Val Pro Gly Arg Gly Arg Ile
210 215 220
ccg tgg gtc gaa atc ggt gaa gca ctg gct gat att ggc tac aac ggt 720
Pro Trp Val Glu Ile Gly Glu Ala Leu Ala Asp Ile Gly Tyr Asn Gly
225 230 235
tca gtg gtt atg gaa ccg ttt gtt cgt atg ggc ggc acc gtc ggc agc 768
Ser Val Val Met Glu Pro Phe Val Arg Met Gly Gly Thr Val Gly Ser
240 245 250 255
aat att aaa gtg tgg cgc gat atc tct aac ggt gca gat gaa aag atg 816
Asn Ile Lys Val Trp Arg Asp Ile Ser Asn Gly Ala Asp Glu Lys Met
260 265 270
ctg gac cgt gaa gct cag gca gct ctg gac ttc tca cgc tac gtg ctg 864
Leu Asp Arg Glu Ala Gln Ala Ala Leu Asp Phe Ser Arg Tyr Val Leu
275 280 285
gaa tgt cat aaa cac tcg taaagatctg gatcc 897
Glu Cys His Lys His Ser
290
<210> 8
<211> 293
<212> PRT
<213> Clostridium cellulolyticum
<400> 8
Met Lys His Gly Ile Tyr Tyr Ala Tyr Trp Glu Gln Glu Trp Glu Ala
1 5 10 15
Asp Tyr Lys Tyr Tyr Ile Glu Lys Val Ala Lys Leu Gly Phe Asp Ile
20 25 30
Leu Glu Ile Ala Ala Ser Pro Leu Pro Phe Tyr Ser Asp Ile Gln Ile
35 40 45
Asn Glu Leu Lys Ala Cys Ala His Gly Asn Gly Ile Thr Leu Thr Val
50 55 60
Gly His Gly Pro Ser Ala Glu Gln Asn Leu Ser Ser Pro Asp Pro Asp
65 70 75 80
Ile Arg Lys Asn Ala Lys Ala Phe Tyr Thr Asp Leu Leu Lys Arg Leu
85 90 95
Tyr Lys Leu Asp Val His Leu Ile Gly Gly Ala Leu Tyr Ser Tyr Trp
100 105 110
Pro Ile Asp Tyr Thr Lys Thr Ile Asp Lys Lys Gly Asp Trp Glu Arg
115 120 125
Ser Val Glu Ser Val Arg Glu Val Ala Lys Val Ala Glu Ala Cys Gly
130 135 140
Val Asp Phe Cys Leu Glu Val Leu Asn Arg Phe Glu Asn Tyr Leu Ile
145 150 155 160
Asn Thr Ala Gln Glu Gly Val Asp Phe Val Lys Gln Val Asp His Asn
165 170 175
Asn Val Lys Val Met Leu Asp Thr Phe His Met Asn Ile Glu Glu Asp
180 185 190
Ser Ile Gly Gly Ala Ile Arg Thr Ala Gly Ser Tyr Leu Gly His Leu
195 200 205
His Thr Gly Glu Cys Asn Arg Lys Val Pro Gly Arg Gly Arg Ile Pro
210 215 220
Trp Val Glu Ile Gly Glu Ala Leu Ala Asp Ile Gly Tyr Asn Gly Ser
225 230 235 240
Val Val Met Glu Pro Phe Val Arg Met Gly Gly Thr Val Gly Ser Asn
245 250 255
Ile Lys Val Trp Arg Asp Ile Ser Asn Gly Ala Asp Glu Lys Met Leu
260 265 270
Asp Arg Glu Ala Gln Ala Ala Leu Asp Phe Ser Arg Tyr Val Leu Glu
275 280 285
Cys His Lys His Ser
290
<210> 9
<211> 885
<212> DNA
<213> Clostridium scindens
<400> 9
aacagaatag gaatatttat gaatttctgg gttaagaact gggatgcaga tcatgtcaag 60
tatattaaaa aggtatccgg ccttggattt gatattctgg aattccaggc ccaggcgctt 120
ctggagatgg ataagagcag gatggatgag gtcaggcagg cggcaaagga caatggaatc 180
gaactgacct acagccttgg gctgaatcct aagtacgatg tcgcaagccc ggatgcaaaa 240
gtcagggaag gcggaatcga atatctgaag cggatcgtgg agcggattgg atacatggaa 300
ggaaaactgc tttccggagt caactatgcc ggctggggaa gcccggacta tatcgtggat 360
gacaaaagcg agatcgtgga gcacagcatc gaaagcgtcc gccaggtcat taagacggca 420
gaagattatg acgtgactta ctgcgtggag gtcgtgaacc ggtttgaggg catcgtgatg 480
aatacggcaa aggaagccat cgagtacgtg aagcagattg acagtgataa gatcggaatc 540
ctgctggata cctatcatat gaacatcgag gaaggctcta taggagacgc catccgatct 600
gtaggcggat atctgaagaa cttccacact ggagagaaca accgggtcgt tccggggaag 660
gggcacctcg actgggatga aatatttgga gcgctccatg atatcgatta tcagggaagg 720
atcgtgtcag agccgttcgt ccagatgggc ggggaagtcg caagagacat caaggtatgg 780
agagatctgg tggaagatcc ttcagaagaa gtgctggatg aggaggcgcg cttccttctg 840
aattttgaaa aggatatgat ccggaagcac tatggcatag cgtaa 885
<210> 10
<211> 867
<212> DNA
<213> Clostridium hylemonae
<400> 10
aaacatggta tctattatgc atactgggaa caagaatggg cggccgacta caagcgctat 60
gttgaaaagg tggcaaagct tgggtttgac attctggaga tcggcgctgg gccgctgccg 120
gaatacgcag agcaggatgt gaaggaactg aagaaatgtg cgcaggacaa tgggatcacg 180
ctgacggccg gatatggtcc gacgttcaac cacaatatcg gttcttcaga cgccggggta 240
agggaagagg cgctggaatg gtataagagg ttatttgaag tgctggcaga gcttgatatc 300
cacctgatcg gaggggcgct ctattcttac tggcctgtcg attttgcaaa cgccgataaa 360
acggaagact ggaagtggag tgtagagggc atgcagaggc tggcgccggc cgcggccaaa 420
tatgacatca acctgggcat ggaagttctg aaccggtttg agagccatat cctgaataca 480
gccgaggaag gtgtgaagtt tgtagaggaa gtcggcatgg acaacgtaaa ggtcatgctg 540
gatacattcc atatgaatat agaagagcaa agcataggcg gcgcgatccg ccgggcagga 600
aaactgctcg ggcatttcca caccggagaa tgcaaccgca tggtgcccgg gaagggacgt 660
attccatggc gtgagatagg ggatgctctc cgtgatatcg gatatgacgg aactgctgta 720
atggagccgt tcgttcgcat gggaggacag gtcggcgctg atatcaaggt gtggagagac 780
ataagccgtg gagcagacga ggcacagctt gacgatgacg cgcgccgtgc gctggagttc 840
cagagatata tgctggagtg gaagtaa 867
<210> 11
<211> 867
<212> DNA
<213> Desmospora sp
<400> 11
aaatacggtg tctatttcgc ttactgggaa gactcgtggg atgtggattt cgagaagtac 60
gtgcggaaag tgaaaaagtt gggcttcgac atcctcgaag tggcggcatt gggtctcgtc 120
aaccttccgg aggagaaact ggagcggctg aaacaactcg ccgaacagca cgatatcatc 180
ctgacggccg ggatcggcct gccaaaggaa tacgatgtct cgtcaactga caaaaaggtg 240
cgccggaacg gcatctcctt catgaagaaa gtgatggacg cgatgcatca ggccggcatc 300
caccggatcg gcggcacggt ctactcgtat tggccggttg actacagttg ctccttcgac 360
aagccggccg taaggaagca cagcatcgaa agcgtcagag agctggcgga gtacgcacgg 420
cagtacaaca tcacactcct catcgaaacg ctcaaccggt ttgagcagtt tctcctgaac 480
gacgcggagg aagcagtcgc ctatgtgaag gaagtggacg agccgaatgt gaaagtcatg 540
ctcgacacat tccacatgaa catcgaggaa gaccacattg ccgatgccat ccgctacacc 600
ggtgaccacc tcggccaact gcacatcggc gaagcgaatc ggaaagtccc gggcaagggt 660
tcgatgcctt ggacagaaat cggacaggcg ctgaaagaca ttcgctacga tggctacgtt 720
gtcatggaac ccttcatcaa aaccggcgga caggtcggcc gggacatcaa gctctggcgc 780
gatctgtcgg gaaatgcgac ggaggaacag ttggaccggg agctggcaga gtcgctggaa 840
tttgtgaaag cggcgttcgg ggagtaa 867
<210> 12
<211> 879
<212> DNA
<213> Clostridium cellulolyticum
<400> 12
aaacatggta tatactacgc atattgggaa caagaatggg aagctgatta caaatactat 60
attgagaagg ttgcaaagct tggttttgat attctagaga ttgcagcttc accgctacct 120
ttttacagtg acattcagat taatgagctc aaggcatgtg cccatggcaa tggaattaca 180
cttacggtag gccatgggcc tagtgcagaa caaaacctgt cttctcccga ccccgatatt 240
cgcaaaaatg ctaaagcttt ttataccgat ttactcaaac gactttacaa gctggatgta 300
catttgatag gtggggcttt atattcttat tggccgatag attacacaaa gacaattgat 360
aaaaaaggcg attgggaacg cagcgttgaa agtgttcgag aagttgctaa ggtggccgaa 420
gcctgtggag tggatttctg cctagaggtt cttaatagat ttgagaatta tttaattaac 480
acagcacaag agggtgtaga ttttgtaaaa caggttgacc ataacaatgt aaaggtaatg 540
cttgatacct tccatatgaa tattgaggaa gatagtatcg gaggtgcaat caggactgcg 600
ggctcttact tgggacattt acacactggc gaatgtaatc gtaaagttcc cggcagagga 660
agaattccat gggtagaaat tggtgaggct cttgctgaca taggttataa cggtagtgtt 720
gttatggaac cttttgttag aatgggcgga actgtcggat ctaatattaa ggtttggcgt 780
gacattagta acggtgcaga tgagaaaatg ctggatagag aagcacaggc cgcacttgat 840
ttctccagat atgtattaga atgtcataaa cactcctga 879
<210> 13
<211> 289
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial variant of the ketose-3-epimerase of Desmospora sp.
8437.
<400> 13
Met Arg Tyr Gly Ile Tyr Tyr Ala Tyr Trp Glu Asp Ser Trp Asp Ala
1 5 10 15
Asp Phe Glu Lys Tyr Val Lys Lys Val Lys Lys Leu Gly Phe Asp Ile
20 25 30
Ile Glu Val Ala Ala Leu Gly Phe Val Asn Leu Pro Glu Glu Lys Leu
35 40 45
Glu Thr Leu Arg Gln Leu Ala Glu Gln His Asp Ile Ile Leu Thr Ala
50 55 60
Gly Tyr Gly Leu Pro Lys Glu Tyr Asn Val Ser Ser Pro Asp Lys Lys
65 70 75 80
Val Arg Arg Asn Gly Ile Ser Phe Met Lys Lys Val Leu Asp Ala Met
85 90 95
His Gln Leu Gly Ile His Arg Ile Gly Gly Thr Val Phe Ser Tyr Trp
100 105 110
Pro Val Asp Tyr Ser Cys Ser Phe Asp Lys Pro Ala Val Arg Lys His
115 120 125
Ala Ile Glu Ser Val Arg Glu Val Ala Glu Tyr Ala Arg Gln Tyr Asn
130 135 140
Ile Thr Leu Ala Ile Glu Val Leu Asn Arg Phe Glu Gln Phe Val Leu
145 150 155 160
Asn Asp Ala Glu Glu Ala Ile Ala Tyr Val Lys Glu Val Gly Glu Pro
165 170 175
Asn Val Lys Val Met Leu Asp Thr Phe His Met Asn Ile Glu Glu Asp
180 185 190
His Phe Ala Asp Ala Ile Arg Tyr Ala Gly Asp Leu Leu Gly Gln Leu
195 200 205
His Ile Gly Glu Ala Asn Arg Lys Val Pro Gly Lys Gly Ser Leu Pro
210 215 220
Trp Thr Glu Ile Gly Gln Ala Leu Lys Asp Ile Arg Tyr Asp Gly Tyr
225 230 235 240
Val Ile Met Glu Pro Phe Val Lys Thr Gly Gly Thr Val Gly Arg Asp
245 250 255
Val Lys Leu Trp Arg Asp Met Ser Gly Asn Ala Thr Glu Glu Gln Leu
260 265 270
Asp Arg Glu Leu Ala Glu Ser Leu Glu Phe Val Arg Ala Ala Phe Gly
275 280 285
Glu
<210> 14
<211> 289
<212> PRT
<213> Agrobacterium tumefaciens
<400> 14
Met Lys His Gly Ile Tyr Tyr Ser Tyr Trp Glu His Glu Trp Ser Ala
1 5 10 15
Lys Phe Gly Pro Tyr Ile Glu Lys Val Ala Lys Leu Gly Phe Asp Ile
20 25 30
Ile Glu Val Ala Ala His His Ile Asn Glu Tyr Ser Asp Ala Glu Leu
35 40 45
Ala Thr Ile Arg Lys Ser Ala Lys Asp Asn Gly Ile Ile Leu Thr Ala
50 55 60
Gly Ile Gly Pro Ser Lys Thr Lys Asn Leu Ser Ser Glu Asp Ala Ala
65 70 75 80
Val Arg Ala Ala Gly Lys Ala Phe Phe Glu Arg Thr Leu Ser Asn Val
85 90 95
Ala Lys Leu Asp Ile His Thr Ile Gly Gly Ala Leu His Ser Tyr Trp
100 105 110
Pro Ile Asp Tyr Ser Gln Pro Val Asp Lys Ala Gly Asp Tyr Ala Arg
115 120 125
Gly Val Glu Gly Ile Asn Gly Ile Ala Asp Phe Ala Asn Asp Leu Gly
130 135 140
Ile Asn Leu Cys Ile Glu Val Leu Asn Arg Phe Glu Asn His Val Leu
145 150 155 160
Asn Thr Ala Ala Glu Gly Val Ala Phe Val Lys Asp Val Gly Lys Asn
165 170 175
Asn Val Lys Val Met Leu Asp Thr Phe His Met Asn Ile Glu Glu Asp
180 185 190
Ser Phe Gly Asp Ala Ile Arg Thr Ala Gly Pro Leu Leu Gly His Phe
195 200 205
His Thr Gly Glu Ser Asn Arg Arg Val Pro Gly Lys Gly Arg Met Pro
210 215 220
Trp His Glu Ile Gly Leu Ala Leu Arg Asp Ile Asn Tyr Thr Gly Ala
225 230 235 240
Val Ile Met Glu Pro Phe Val Lys Thr Gly Gly Thr Ile Gly Ser Asp
245 250 255
Ile Lys Val Trp Arg Asp Leu Ser Gly Gly Ala Asp Ile Ala Lys Met
260 265 270
Asp Glu Asp Ala Arg Asn Ala Leu Ala Phe Ser Arg Phe Val Leu Gly
275 280 285
Gly
<210> 15
<211> 290
<212> PRT
<213> Pseudomonas cichorii
<400> 15
Met Asn Lys Val Gly Met Phe Tyr Thr Tyr Trp Ser Thr Glu Trp Met
1 5 10 15
Val Asp Phe Pro Ala Thr Ala Lys Arg Ile Ala Gly Leu Gly Phe Asp
20 25 30
Leu Met Glu Ile Ser Leu Gly Glu Phe His Asn Leu Ser Asp Ala Lys
35 40 45
Lys Arg Glu Leu Lys Ala Val Ala Asp Asp Leu Gly Leu Thr Val Met
50 55 60
Cys Cys Ile Gly Leu Lys Ser Glu Tyr Asp Phe Ala Ser Pro Asp Lys
65 70 75 80
Ser Val Arg Asp Ala Gly Thr Glu Tyr Val Lys Arg Leu Leu Asp Asp
85 90 95
Cys His Leu Leu Gly Ala Pro Val Phe Ala Gly Leu Thr Phe Cys Ala
100 105 110
Trp Pro Gln Ser Pro Pro Leu Asp Met Lys Asp Lys Arg Pro Tyr Val
115 120 125
Asp Arg Ala Ile Glu Ser Val Arg Arg Val Ile Lys Val Ala Glu Asp
130 135 140
Tyr Gly Ile Ile Tyr Ala Leu Glu Val Val Asn Arg Phe Glu Gln Trp
145 150 155 160
Leu Cys Asn Asp Ala Lys Glu Ala Ile Ala Phe Ala Asp Ala Val Asp
165 170 175
Ser Pro Ala Cys Lys Val Gln Leu Asp Thr Phe His Met Asn Ile Glu
180 185 190
Glu Thr Ser Phe Arg Asp Ala Ile Leu Ala Cys Lys Gly Lys Met Gly
195 200 205
His Phe His Leu Gly Glu Ala Asn Arg Leu Pro Pro Gly Glu Gly Arg
210 215 220
Leu Pro Trp Asp Glu Ile Phe Gly Ala Leu Lys Glu Ile Gly Tyr Asp
225 230 235 240
Gly Thr Ile Val Met Glu Pro Phe Met Arg Lys Gly Gly Ser Val Ser
245 250 255
Arg Ala Val Gly Val Trp Arg Asp Met Ser Asn Gly Ala Thr Asp Glu
260 265 270
Glu Met Asp Glu Arg Ala Arg Arg Ser Leu Gln Phe Val Arg Asp Lys
275 280 285
Leu Ala
290
Claims (27)
- 단백질로서,
SEQ ID NO: 6에 대하여 적어도 89%의 서열 동일성을 가지는 폴리펩타이드 서열을 포함하고,
상기 단백질은 사이코스-3-에피메라아제 활성을 가지고,
상기 폴레핍티드 서열은 SEQ ID NO: 6과 동일하지 않은 것인 단백질. - 청구항 1에 있어서,
상기 폴리펩타이드 서열은, SEQ ID NO: 6에 대하여 적어도 90%, 95% 또는 99%의 서열 동일성을 가지는 단백질. - 청구항 1에 있어서,
상기 단백질은, 고체기질 상에 고정화되는 단백질. - SEQ ID NO: 6에 대하여 적어도 89%의 서열 동일성을 가지는 폴리펩타이드 서열을 포함하는 단백질을 포함하는, 사이코스 합성을 위한 조성물.
- SEQ ID NO: 6에 대하여 적어도 89%의 서열 동일성을 가지는 폴리펩타이드 서열을 포함하는 단백질을 인코드하는 폴리뉴클레오타이드 서열을 포함하는 핵산분자로서,
상기 폴리펩타이드 서열은 SEQ ID NO: 6과 동일하지 않은 것인 핵산분자. - 청구항 5에 있어서,
i) SEQ ID NO: 5에 대하여 적어도 89%, 90%, 95% 또는 99%의 서열 동일성을 가지거나; 또는
ii) 엄격한 조건하에 있어서, SEQ ID NO: 5에 규정된 서열에 대하여 상보적인 서열을 가지는 폴리뉴클레오타이드에 혼성화하는
폴리뉴클레오타이드 서열을 포함하는 핵산분자. - 청구항 5 또는 청구항 6에 있어서,
상기 폴리뉴클레오타이드 서열은 SEQ ID NO: 5의 핵산 서열을 포함하지 않는 핵산분자. - 청구항 5 또는 청구항 6에 따른 핵산분자를 포함하는 벡터.
- 청구항 5 또는 청구항 6에 따른 재조합 핵산분자를 포함하는 숙주세포.
- 청구항 9에 있어서,
상기 숙주세포는, 효모, 박테리아 또는 그 이외의 미생물이거나 혹은 포유동물, 식물 또는 그 이외의 세포 배양물인 숙주세포. - 청구항 10에 있어서,
상기 숙주세포는 대장균인 숙주세포. - 사이코스의 생성방법으로서,
과당 기질을 사이코스로 전환하는 조건 하에 있어서, 단백질을 과당 기질과 접촉시키는 단계를 포함하고,
상기 단백질은 SEQ ID NO: 6에 대하여 적어도 89%의 서열 동일성을 가지는 폴리펩타이드 서열을 포함하고, 상기 단백질은 사이코스-3-에피메라아제 활성을 갖는 것인 방법. - 청구항 12에 있어서,
상기 단백질은, 숙주세포 내에 존재하는 방법. - 청구항 12에 있어서,
상기 단백질은, 단리 형태인 방법. - 청구항 12 내지 청구항 14 중 어느 한 항에 있어서,
상기 조건에는, 상기 단백질 및 상기 과당 기질을 25℃~75℃의 온도 범위로 유지하는 것이 포함되는 방법. - 청구항 12 내지 청구항 14 중 어느 한 항에 있어서,
상기 조건에는, 상기 단백질 및 상기 과당 기질을 pH4~pH10의 범위로 유지하는 것이 포함되는 방법. - 청구항 12 내지 청구항 14 중 어느 한 항에 있어서,
상기 조건에는, 상기 과당 기질을 75%~95%(w/v)의 농도 범위로 유지하는 것이 포함되는 방법. - 핵산분자로서,
i) SEQ ID NO: 5에 대하여 적어도 89%의 서열 동일성을 가지거나; 또는
ii) 엄격한 조건하에 있어서, SEQ ID NO: 5에 규정된 서열에 대하여 상보적인 서열을 가지는 폴리뉴클레오타이드에 혼성화하는
폴리뉴클레오타이드 서열을 포함하는 핵산분자. - 사이코스 3-에피메라아제 활성을 가지는 폴리펩타이드를 인코드하는 폴리뉴클레오타이드 서열을 포함한 재조합 핵산분자를 포함하는 숙주세포로서,
상기 폴리뉴클레오타이드 서열은,
i) SEQ ID NO: 5에 대하여 적어도 89%의 서열 동일성을 가지거나; 또는
ii) 엄격한 조건하에 있어서, SEQ ID NO: 5에 규정된 서열에 대하여 상보적인 서열을 가지는 폴리뉴클레오타이드에 혼성화하는 것인,
숙주세포. - 사이코스 3-에피메라아제 활성을 가지는 폴리펩타이드를 인코드하는 폴리뉴클레오타이드를 포함한 핵산분자를 포함하는 벡터로서,
상기 폴리뉴클레오타이드 서열은,
i) SEQ ID NO: 5에 대하여 적어도 89%의 서열 동일성을 가지거나; 또는
ii) 엄격한 조건하에 있어서, SEQ ID NO: 5에 규정된 서열에 대하여 상보적인 서열을 가지는 폴리뉴클레오타이드에 혼성화하는 것인,
벡터. - i) 사이코스 3-에피메라아제 활성을 가지는 단백질을 인코드하는 폴리뉴클레오타이드 서열을 가지는 핵산분자를 포함한 벡터를 제공하는 단계로서, 상기 폴리뉴클레오타이드 서열은, a) SEQ ID NO: 5에 대하여 적어도 89%의 서열 동일성을 가지거나; 또는 b) 엄격한 조건하에 있어서, SEQ ID NO: 5에 규정된 서열에 대하여 상보적인 서열을 가지는 폴리뉴클레오타이드에 혼성화하는 것인 단계;
ii) 상기 폴리뉴클레오타이드 서열에 인코드된 사이코스 3-에피메라아제 활성을 가지는 상기 단백질을 합성하는 단계;
iii) 과당을 사이코스 3-에피메라아제 활성을 가지는 상기 단백질에 접촉시켜, 과당으로부터 사이코스로의 전환을 허용하는 조건 하에, 상기 과당 및 상기 단백질을 유지하는 단계; 및
iv) 상기 단계 iii)에서 생성된 사이코스를 적어도 부분적으로 정제하는 단계를 포함하는, 사이코스의 생성방법. - 청구항 21에 있어서,
상기 핵산 서열은, SEQ ID NO: 5에 대하여 적어도 95% 또는 99%의 서열 동일성을 가지거나 혹은 100%의 서열 동일성을 가지는 것인 방법. - 청구항 21에 있어서,
상기 단계 ii)는 컴피턴트 숙주세포를 상기 단계 i)의 벡터로 형질전환시키고 및, 선택적으로, 상기 형질전환된 숙주세포를 배양하는 것을 포함하는 방법. - 청구항 23에 있어서,
상기 단계 iii)은 상기 형질전환된 세포를 과당과 혼합하는 것을 포함하는 방법. - 청구항 23에 있어서,
상기 단계 iii)은 상기 숙주세포로부터 사이코스 3-에피메라아제 활성을 가지는 단백질을 단리시키고 및 상기 단백질을 상기 과당과 혼합하는 것을 포함하는 방법. - 청구항 25에 있어서,
상기 사이코스 3-에피메라아제 활성을 가지는 단백질은 상기 단백질의 단리 후에 기질상에 고정화되는 방법. - 청구항 21에 있어서, 상기 단계 ii)에서, 상기 사이코스 3-에피메라아제 활성을 가지는 단백질을 인 비트로 번역으로 합성하고 및 후속하여 단리시키는 방법.
Applications Claiming Priority (5)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201261706338P | 2012-09-27 | 2012-09-27 | |
| US61/706,338 | 2012-09-27 | ||
| GB1220554.8 | 2012-11-15 | ||
| GB1220554.8A GB2508586B (en) | 2012-09-27 | 2012-11-15 | A protein |
| PCT/GB2013/052531 WO2014049373A1 (en) | 2012-09-27 | 2013-09-27 | 3-epimerase |
Related Parent Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020157009700A Division KR20150059760A (ko) | 2012-09-27 | 2013-09-27 | 3-에피메라아제 |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20200028039A true KR20200028039A (ko) | 2020-03-13 |
Family
ID=47521205
Family Applications (2)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020157009700A KR20150059760A (ko) | 2012-09-27 | 2013-09-27 | 3-에피메라아제 |
| KR1020207006415A KR20200028039A (ko) | 2012-09-27 | 2013-09-27 | 3-에피메라아제 |
Family Applications Before (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020157009700A KR20150059760A (ko) | 2012-09-27 | 2013-09-27 | 3-에피메라아제 |
Country Status (16)
| Country | Link |
|---|---|
| US (4) | US9725707B2 (ko) |
| EP (2) | EP2900827B1 (ko) |
| JP (1) | JP6340005B2 (ko) |
| KR (2) | KR20150059760A (ko) |
| CN (1) | CN104903457A (ko) |
| AR (1) | AR092712A1 (ko) |
| AU (2) | AU2013322364B2 (ko) |
| CA (1) | CA2886514C (ko) |
| CL (1) | CL2015000757A1 (ko) |
| GB (2) | GB2583417B (ko) |
| IL (1) | IL237900B (ko) |
| IN (1) | IN2015DN03196A (ko) |
| MX (2) | MX2015004060A (ko) |
| PL (1) | PL2900827T3 (ko) |
| TW (1) | TWI675916B (ko) |
| WO (1) | WO2014049373A1 (ko) |
Families Citing this family (22)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| GB2583417B (en) | 2012-09-27 | 2021-03-31 | Tate & Lyle Ingredients Americas Llc | A protein |
| KR101473918B1 (ko) * | 2014-05-28 | 2014-12-17 | 대상 주식회사 | 사이코스 에피머화 효소, 이의 제조방법 및 이를 이용한 사이코스의 제조방법 |
| KR101577147B1 (ko) * | 2014-10-01 | 2015-12-11 | 경상대학교산학협력단 | 사이코스의 생산 방법 |
| WO2016160573A1 (en) * | 2015-03-27 | 2016-10-06 | Archer Daniels Midland Co. | Fructose to allulose conversion |
| HUE058347T2 (hu) | 2015-05-22 | 2022-07-28 | Archer Daniels Midland Co | Epimeráz enzimek alkalmazása fruktóz allulózzá történõ átalakítására |
| KR101785300B1 (ko) * | 2015-12-23 | 2017-11-15 | 대상 주식회사 | 미생물 균체 고정화 장치 및 이를 이용한 미생물 균체 고정화 방법 |
| CN106148311B (zh) * | 2016-09-12 | 2019-06-21 | 上海立足生物科技有限公司 | 一种d-阿洛酮糖-3-差向异构酶的突变体及其应用 |
| JP6990656B2 (ja) * | 2016-09-14 | 2022-02-03 | 松谷化学工業株式会社 | 固定化アルロースエピメラーゼの製造方法 |
| BR112019007257A2 (pt) * | 2016-10-11 | 2019-07-02 | Braskem Sa | micro-organismos e métodos para a coprodução de etileno glicol e isobuteno |
| WO2018081557A2 (en) | 2016-10-28 | 2018-05-03 | Tate & Lyle Ingredients Americas Llc | Method for producing allulose crystals |
| KR102065155B1 (ko) * | 2016-12-08 | 2020-02-11 | 주식회사 삼양사 | 사이코스의 제조방법 |
| PE20191355A1 (es) * | 2016-12-14 | 2019-10-01 | Bonumose Llc | Produccion enzimatica de d-alulosa |
| CN108239632B (zh) * | 2016-12-26 | 2023-05-05 | 上海立足生物科技有限公司 | 一种热稳定性得到改善的d-阿洛酮糖-3-差向异构酶的突变体及其应用 |
| BR112019013853A2 (pt) * | 2017-01-06 | 2020-01-28 | Greenlight Biosciences Inc | produção de açúcares isentos de células |
| KR101981430B1 (ko) | 2017-06-23 | 2019-05-23 | 씨제이제일제당 (주) | D-사이코스 붕산염 착물로부터 크로마토그래피를 이용한 d-사이코스의 생산 방법 및 d-사이코스를 포함하는 조성물 |
| US11377650B2 (en) * | 2017-08-31 | 2022-07-05 | Novozymes A/S | Polypeptides having D-psicose 3 epimerase activity and polynucleotides encoding same |
| CN111836889B (zh) * | 2018-01-24 | 2024-02-09 | 松谷化学工业株式会社 | 热稳定性提高的酮糖3-差向异构酶 |
| WO2019166514A1 (en) | 2018-02-28 | 2019-09-06 | C-Lecta Gmbh | Enzymatic in-situ fortification of food with functional carbohydrates |
| EP3833756B9 (en) | 2018-08-08 | 2023-10-04 | Archer Daniels Midland Company | Epimerase enzymes and their use |
| CN110862980B (zh) * | 2019-11-29 | 2021-05-04 | 浙江工业大学 | 一种d-阿洛酮糖3-差向异构酶突变体及其应用 |
| EP4000419A1 (de) | 2020-11-23 | 2022-05-25 | Savanna Ingredients GmbH | Trocknung von allulosekristallen |
| CN114790469B (zh) * | 2021-01-26 | 2024-03-26 | 中国科学院天津工业生物技术研究所 | 一种酶促合成阿洛酮糖的方法 |
Citations (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| KR20090098938A (ko) | 2008-03-12 | 2009-09-18 | 주식회사 케이엠더블유 | 무선통신기기의 함체장치 |
Family Cites Families (20)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| JP3333969B2 (ja) | 1992-10-08 | 2002-10-15 | 株式会社林原生物化学研究所 | D−ケトヘキソース・3−エピメラーゼとその製造方法並びに用途 |
| KR100744479B1 (ko) | 2005-06-01 | 2007-08-01 | 씨제이 주식회사 | 사이코스 에피머화 효소에 의한 사이코스의 생산 방법 |
| GB0512150D0 (en) | 2005-06-15 | 2005-07-20 | Scottish Crop Res Inst | Production of L-ascorbic acid |
| EP1956088B1 (en) | 2005-11-15 | 2013-10-23 | Hayashibara Co., Ltd. | Ketose 3-epimerase, process for production thereof, and use thereof |
| US8940350B2 (en) | 2005-11-23 | 2015-01-27 | The Coca-Cola Company | Cereal compositions comprising high-potency sweeteners |
| JP4942001B2 (ja) | 2006-11-10 | 2012-05-30 | 松谷化学工業株式会社 | D−プシコース含有甘味料およびそれを使用して得られた飲食品など |
| KR100832339B1 (ko) | 2006-12-11 | 2008-05-26 | 솔젠트 (주) | 과당을 사이코스로 전환하는 신규한 시노리조비움 속균주와 이를 이용한 사이코스 생산법 |
| KR20100018568A (ko) * | 2007-05-18 | 2010-02-17 | 마쓰다니가가꾸고오교가부시끼가이샤 | 슈크로오스성 감미질을 가지는 신규 감미료, 그 제조법 및 용도 |
| CN100577793C (zh) | 2007-12-12 | 2010-01-06 | 江南大学 | 微生物转化d-果糖制备d-阿洛酮糖的菌种和方法 |
| KR20110035805A (ko) * | 2009-09-30 | 2011-04-06 | 씨제이제일제당 (주) | 사이코스-에피머화 효소의 고정화 및 이를 이용한 사이코스의 제조방법 |
| KR101106253B1 (ko) | 2009-10-16 | 2012-01-18 | 경상대학교산학협력단 | 사이코스 3-에피머라제 효소를 코딩하는 폴리뉴클레오티드를 포함하는 대장균 및 그를 이용하여 사이코스를 생산하는 방법 |
| CN102373230A (zh) * | 2010-08-27 | 2012-03-14 | 天津工业生物技术研究所 | 某种梭菌d-塔格糖3-差向异构酶的核苷酸序列及其应用 |
| KR101944027B1 (ko) | 2011-07-06 | 2019-01-30 | 마쓰다니가가꾸고오교가부시끼가이샤 | 아스로박터 글로비포미스가 생산하는 효소 |
| CN103131721B (zh) | 2011-11-25 | 2014-10-29 | 中国科学院天津工业生物技术研究所 | 瘤胃菌d-塔格糖3-差向异构酶的核苷酸序列及其应用 |
| WO2013126721A1 (en) * | 2012-02-23 | 2013-08-29 | Massachusetts Institute Of Technology | Engineering microbes and metabolic pathways for the production of ethylene glycol |
| WO2014025235A1 (ko) | 2012-08-10 | 2014-02-13 | 주식회사 삼양제넥스 | 사이코스 에피머화 효소 및 이를 이용한 사이코스로 전환용 조성물 |
| GB2583417B (en) * | 2012-09-27 | 2021-03-31 | Tate & Lyle Ingredients Americas Llc | A protein |
| GB201309076D0 (en) | 2013-03-15 | 2013-07-03 | Tate & Lyle Ingredients | Improved sweetener |
| GB201309079D0 (en) | 2013-03-15 | 2013-07-03 | Tate & Lyle Ingredients | Improved sweetner |
| GB201309077D0 (en) | 2013-03-15 | 2013-07-03 | Tate & Lyle Ingredients | Improved sweetener |
-
2012
- 2012-11-15 GB GB2007535.4A patent/GB2583417B/en active Active
- 2012-11-15 GB GB1220554.8A patent/GB2508586B/en active Active
-
2013
- 2013-09-27 MX MX2015004060A patent/MX2015004060A/es unknown
- 2013-09-27 KR KR1020157009700A patent/KR20150059760A/ko active Application Filing
- 2013-09-27 KR KR1020207006415A patent/KR20200028039A/ko not_active Application Discontinuation
- 2013-09-27 US US14/431,549 patent/US9725707B2/en active Active
- 2013-09-27 TW TW102135138A patent/TWI675916B/zh active
- 2013-09-27 JP JP2015533696A patent/JP6340005B2/ja active Active
- 2013-09-27 AU AU2013322364A patent/AU2013322364B2/en active Active
- 2013-09-27 WO PCT/GB2013/052531 patent/WO2014049373A1/en active Application Filing
- 2013-09-27 PL PL13773831T patent/PL2900827T3/pl unknown
- 2013-09-27 CN CN201380050744.8A patent/CN104903457A/zh active Pending
- 2013-09-27 AR ARP130103485A patent/AR092712A1/es unknown
- 2013-09-27 EP EP13773831.6A patent/EP2900827B1/en active Active
- 2013-09-27 CA CA2886514A patent/CA2886514C/en active Active
- 2013-09-27 EP EP18150796.3A patent/EP3336194B1/en active Active
- 2013-09-27 IN IN3196DEN2015 patent/IN2015DN03196A/en unknown
-
2015
- 2015-03-23 IL IL237900A patent/IL237900B/en active IP Right Grant
- 2015-03-25 CL CL2015000757A patent/CL2015000757A1/es unknown
- 2015-03-27 MX MX2020010399A patent/MX2020010399A/es unknown
-
2017
- 2017-03-21 AU AU2017201896A patent/AU2017201896A1/en not_active Abandoned
- 2017-06-30 US US15/639,214 patent/US10294469B2/en active Active
-
2019
- 2019-03-29 US US16/370,163 patent/US20190218541A1/en not_active Abandoned
-
2020
- 2020-12-31 US US17/139,605 patent/US11859224B2/en active Active
Patent Citations (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| KR20090098938A (ko) | 2008-03-12 | 2009-09-18 | 주식회사 케이엠더블유 | 무선통신기기의 함체장치 |
Non-Patent Citations (3)
| Title |
|---|
| 미국 공개특허공보 제2011/0275138호는, 리조비움속(Rhizobium genus)의 미생물 유래의 케토오스-3-에피메라아제를 개시하고 있다. 이 단백질은, D-케토펜토오스 또는 L-케토펜토오스 및 D-케토헥소오스 또는 L-케토헥소오스에 높은 특이성을 나타내며, 특히 D-과당 및 D-사이코스에 높은 특이성을 나타낸다. 이 문서는 또한, 단백질을 이용하여 케토오스를 생성하는 프로세스를 개시하고 있다. |
| 케토오스-3-에피메라아제는, 과당 및 알룰로오스를 상호 전환할 수 있다. 미국 특허공보 제8,030,035호 및 PCT 공개공보 WO2011/040708호는, 아그로박테리움투메파시엔스(Agrobacterium tumefaciens) 유래이고, 또한, 사이코스(psicose)-3-에피메라아제 활성을 가지는 단백질을 D-과당과 반응시킴으로써, D-사이코스(알룰로오스의 대체명)를 생성할 수 있는 것을 개시하고 있다. |
| 한국 특허공보 제100832339호는, 과당을 사이코스(즉, 알룰로오스)로 전환할 수 있는 시노리조비움(Sinorhizobium) YB-58형과, 시노리조비움 YB-58형의 균체를 이용한 사이코스의 생성방법을 개시하고 있다. |
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| US11859224B2 (en) | Methods for manufacturing a product using a 3-epimerase | |
| EP2470668B1 (en) | Immobilization of psicose-epimerase and a method of producing d-psicose using the same | |
| JP5643756B2 (ja) | バチルス・サーキュランス由来のβ−ガラクトシダーゼ | |
| CN112063666B (zh) | 一种重组蔗糖异构酶在转化蔗糖制备异麦芽酮糖中的应用 | |
| US20080124771A1 (en) | Food Grade Thermophilic Arabinose Isomerase Expressed from Gras, and Tagatose Manufacturing Method By Using It | |
| CN113462678B (zh) | 一种谷氨酸脱羧酶突变体 | |
| CA2909440A1 (en) | A method of production of rare disaccharides | |
| JP3650632B2 (ja) | マルトースをトレハロースに変換する組換え型酵素 | |
| US8137946B2 (en) | Recombinant GRAS strains expressing thermophilic arabinose isomerase as an active form and method of preparing food grade tagatose by using the same | |
| CN110872586B (zh) | 固定化葡萄糖基转移酶及制备方法及催化生产莱鲍迪苷d的方法 | |
| KR102312807B1 (ko) | 비피도박테리움 속의 저항전분 분해 활성을 가지는 아밀라아제들 및 이의 용도 | |
| KR101237857B1 (ko) | 신규한 알파-엘-아라비노퓨라노시데이즈 및 그 효소를 이용한 반섬유소 아라비난으로부터 아라비노스의 제조 방법 | |
| KR20200047176A (ko) | 투라노스의 제조방법 | |
| CN113817701A (zh) | α-1,2-岩藻糖基转移酶突变体及其应用 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A107 | Divisional application of patent | ||
| A201 | Request for examination | ||
| E902 | Notification of reason for refusal | ||
| E601 | Decision to refuse application | ||
| J201 | Request for trial against refusal decision | ||
| J301 | Trial decision |
Free format text: TRIAL NUMBER: 2020101003148; TRIAL DECISION FOR APPEAL AGAINST DECISION TO DECLINE REFUSAL REQUESTED 20201224 Effective date: 20220224 |