KR101770429B1 - 인간 c-fms 항원 결합 단백질 - Google Patents
인간 c-fms 항원 결합 단백질 Download PDFInfo
- Publication number
- KR101770429B1 KR101770429B1 KR1020107005166A KR20107005166A KR101770429B1 KR 101770429 B1 KR101770429 B1 KR 101770429B1 KR 1020107005166 A KR1020107005166 A KR 1020107005166A KR 20107005166 A KR20107005166 A KR 20107005166A KR 101770429 B1 KR101770429 B1 KR 101770429B1
- Authority
- KR
- South Korea
- Prior art keywords
- comprises seq
- group
- seq
- antigen binding
- delete delete
- Prior art date
Links
- XDTMQSROBMDMFD-UHFFFAOYSA-N C1CCCCC1 Chemical compound C1CCCCC1 XDTMQSROBMDMFD-UHFFFAOYSA-N 0.000 description 1
- SLLWNTCCCOFMBU-UHFFFAOYSA-N CCC1(C)C(CCCCN)C1 Chemical compound CCC1(C)C(CCCCN)C1 SLLWNTCCCOFMBU-UHFFFAOYSA-N 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/395—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P1/00—Drugs for disorders of the alimentary tract or the digestive system
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P17/00—Drugs for dermatological disorders
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P17/00—Drugs for dermatological disorders
- A61P17/06—Antipsoriatics
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P19/00—Drugs for skeletal disorders
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P19/00—Drugs for skeletal disorders
- A61P19/02—Drugs for skeletal disorders for joint disorders, e.g. arthritis, arthrosis
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P19/00—Drugs for skeletal disorders
- A61P19/08—Drugs for skeletal disorders for bone diseases, e.g. rachitism, Paget's disease
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P25/00—Drugs for disorders of the nervous system
- A61P25/28—Drugs for disorders of the nervous system for treating neurodegenerative disorders of the central nervous system, e.g. nootropic agents, cognition enhancers, drugs for treating Alzheimer's disease or other forms of dementia
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P29/00—Non-central analgesic, antipyretic or antiinflammatory agents, e.g. antirheumatic agents; Non-steroidal antiinflammatory drugs [NSAID]
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P3/00—Drugs for disorders of the metabolism
- A61P3/08—Drugs for disorders of the metabolism for glucose homeostasis
- A61P3/10—Drugs for disorders of the metabolism for glucose homeostasis for hyperglycaemia, e.g. antidiabetics
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
- A61P35/02—Antineoplastic agents specific for leukemia
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P37/00—Drugs for immunological or allergic disorders
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P37/00—Drugs for immunological or allergic disorders
- A61P37/02—Immunomodulators
- A61P37/06—Immunosuppressants, e.g. drugs for graft rejection
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P43/00—Drugs for specific purposes, not provided for in groups A61P1/00-A61P41/00
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/705—Receptors; Cell surface antigens; Cell surface determinants
- C07K14/715—Receptors; Cell surface antigens; Cell surface determinants for cytokines; for lymphokines; for interferons
- C07K14/7153—Receptors; Cell surface antigens; Cell surface determinants for cytokines; for lymphokines; for interferons for colony-stimulating factors [CSF]
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/2866—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against receptors for cytokines, lymphokines, interferons
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/505—Medicinal preparations containing antigens or antibodies comprising antibodies
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/20—Immunoglobulins specific features characterized by taxonomic origin
- C07K2317/21—Immunoglobulins specific features characterized by taxonomic origin from primates, e.g. man
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/30—Immunoglobulins specific features characterized by aspects of specificity or valency
- C07K2317/33—Crossreactivity, e.g. for species or epitope, or lack of said crossreactivity
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/30—Immunoglobulins specific features characterized by aspects of specificity or valency
- C07K2317/34—Identification of a linear epitope shorter than 20 amino acid residues or of a conformational epitope defined by amino acid residues
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/40—Immunoglobulins specific features characterized by post-translational modification
- C07K2317/41—Glycosylation, sialylation, or fucosylation
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/56—Immunoglobulins specific features characterized by immunoglobulin fragments variable (Fv) region, i.e. VH and/or VL
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/56—Immunoglobulins specific features characterized by immunoglobulin fragments variable (Fv) region, i.e. VH and/or VL
- C07K2317/565—Complementarity determining region [CDR]
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/56—Immunoglobulins specific features characterized by immunoglobulin fragments variable (Fv) region, i.e. VH and/or VL
- C07K2317/567—Framework region [FR]
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/73—Inducing cell death, e.g. apoptosis, necrosis or inhibition of cell proliferation
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/76—Antagonist effect on antigen, e.g. neutralization or inhibition of binding
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/90—Immunoglobulins specific features characterized by (pharmaco)kinetic aspects or by stability of the immunoglobulin
- C07K2317/92—Affinity (KD), association rate (Ka), dissociation rate (Kd) or EC50 value
Abstract
인간 c-fms 단백질과 결합하는 항원 결합 단백질들이 제공된다. 상기 항원 결합 단백질을 인코딩하는 핵산들, 이들을 인코딩하는 벡터들 및 세포들이 또한 제공된다. 상기 항원 결합 단백질들은 c-fms의 CSF-1에로의 결합을 저해하고, 종양들 내로의 단핵구 이동을 감소시키고, 그리고 종양-연관 마크로파지들의 축적을 감소시킬 수 있다.
Description
연관된 출원들의 교차 참조
본 출원은 2007년 8월 21일자로 출원된 미합중국 가특허출원 제60/957,148호 및 2008년 7월 29일자로 출원된 미합중국 가특허출원 제61/084,588호의 우선권을 주장하며, 이들을 전 목적들에 대하여 그들 전체를 본 명세서에 참조로 인용한다.
본 발명은 인간 C-FMS 항원 결합 단백질에 관한 것이다.
많은 인간 및 생쥐 종양세포주(tumor cell lines)들은 사이토카인 콜로니자극인자-1(CSF-1 ; Colony Stimulating Factor-1, 또한 마크로파지-콜로니자극인자, M-CSF라고도 알려짐)를 분비하고, 이는 차례로 상기 수용체 콜로니-펠린 맥도노우 균주(c-fms ; colony-Feline McDonough Strain))를 통하여 단핵구/대식세포들을 유인하고, 그 생존을 촉진시키고, 그리고 활성화시킨다. 종양 연관 마크로파지(TAMs ; tumor associated macrophages ; 또한 종양 침윤성 마크로파지(TIMs ; tumor infiltrating macrophages)로 알려지기도 함)가 세포종(cell tumor) 인구의 50% 정도를 포함하는 종양 기질(tumor stroma)의 주요 구성요소가 될 수 있다. 켈리와 그의 동료들(Kelly et al)의 1988, Br. J. Cancer 57:174-177; 리크와 그의 동료들(Leek et al)의 1994, J. Leukoc. Biol. 56:423- 435을 참조하시오. 원발성 인간 종양들의 조사들에 있어서, CSF-1 mRNA 발현에 대한 광범위한 증거들이 있다. 게다가, 많은 연구들에서 상승된 혈청 CSF-1, TAMs의 수, 또는 조직 CSF-1 및/또는 c-fms의 존재들이 암환자들의 형편없는 예후(prognosis)와 연관이 있다는 것이 입증되었다.
TAMs가 PDGF, TGF-β 및 EGF의 분비를 통한 종양 세포들에 대한 직접적인 미토겐 활성(mitogenic activity) 및 ECM-분해효소(ECM-degrading enzymes)들의 생산을 통한 전이(metastasis)를 포함하여 다양한 수단들에 의한 종양 성장, 전이 및 생존을 유지한다(리크와 해리스(Leek and Harris)의 2002, J. Mammary Gland Biol and Neoplasia 7:177-189; 및 루이스와 폴라드(Lewis and Pollard)의 2006, CancerRes 66:605-612)에서 리뷰됨). TAMs에 의한 종양 지지의 다른 중요한 수단은 COX-2, VEGFs, FGFs, EGF, 산화질소(nitric oxide), 안지오포이에틴(angiopoietins) 및 MMPs 등과 같은 다양한 전혈관형성인자(proangiogenic factors)의 생산을 통한 종양들의 신혈관형성(neo-vascularization)에의 기여이다. 드라노프와 그의 동료들(Dranoff et al.)의 2004, Nat. Rev. Cancer 4: 11-22; 맥믹킹과 그의 동료들(MacMicking et al.)의 1997, Annu. Rev. Immunol. 15:323-350; 만토바니와 그의 동료들(Mantovani et al.)의 1992, Immunol. Today 13:265-270들을 참조하시오. 게다가, CSF-1-유도 마크로파지들은 프로스타글란딘(prostaglandins), 인돌아민 2,3 디옥시게나아제(indolamine 2,3 dioxigenase), 산화질소, IL-10 및 TGFβ 등과 같은 여러 인자들의 생산을 통하여 면역억제성(immunosuppressive)이 될 수 있다. 맥믹킹과 그의 동료들의 1997, Annu. Rev. Immunol. 15:323-350; 브론테와 그의 동료들(Bronte et al)의 2001, J. Immunother. 24:431-446들을 참조하시오.
CSF-1은 막-결합으로서 및 가용성 사이토카인(soluble cytokine)으로서의 둘 다로 발현되고(쎄레티와 그의 동료들(Cerretti et al)의 1988, Mol. Immunol. 25:761-770; 도빈과 그의 동료들(Dobbin et al)의 2005, Bioinformatics 21:2430-2437; 왕과 그의 동료들(Wong et al)의 1987, Biochem. Pharmacol. 36:4325-4329) 그리고 마크로파지들과 그들의 전구체들의 생존, 증식, 주화성(chemotaxis) 및 활성을 조절한다(보우레테와 그의 동료들(Bourette et al)의 2000, Growth Factors 17:155-166; 쎄크치니와 그의 동료들(Cecchini et al.)의 1994, Development 120:1357-1372; Hamilton, 1997, J. Leukoc. Biol. 62:145-155; Hume, 1985, Sci. Prog. 69:485-494; Sasmono and Hume, in: The innate immune response to infection (eds. Kaufmann, S., Gordon, S. & Medzhitov, R.) 71-94 (ASM Press, New York, 2004); 로스와 오우거(Ross and Auger), in: The macrophage (eds. Burke, B. & Lewis, C.) (Oxford University Press, Oxford, 2002)).
c-fms 원발암유전자(proto-oncogene ; 또한 M-CSFR, CSF-IR 또는 CDl 15로 알려진)인 동족수용체(cognate receptor)는 연관된 티로신키나아제 활성(tyrosine kinase activity)을 갖는 165-kD 당단백질(glycoprotein)이며, PDGFR-α, PDGFR-β, VEGFRl, VEGFR2, VEGFR3, Flt3 및 c-kit들을 포함하는 클래스3 수용체(class Ⅲ receptor) 티로신키나아제족에 속한다. 블룸-얀센과 헌터(Blume-Jensen and Hunter)의 2001, Nature 411:355-365; 슐레싱거와 울리치(Schlessinger and Ullrich)의 1992, Neuron 9:383-391; 쉐르와 그의 동료들(Sherr et al)의 1985 Cell 41:665-676; 반 데르 기어와 그의 동료들(van der Geer et al)의 1994, Annu. Rev. Cell. Biol. 10:251-337들을 참조하시오. 펠린육종 바이러스(feline sarcoma virus)의 맥도노우 균주(McDonough strain)에 의해 수송되는 c-fms, v-fms 발암 형태는 필수적으로 활성화되는 프로테인 키나아제 활성(protein kinase activity)을 부여하도록 돌연변이된다(쉐르와 그의 동료들의 1985, Cell 41:665-676; 로우셀과 쉐르(Roussel and Sherr)의 2003, Cell Cycle 2: 5-6). 정상 세포들에서의 c-fms의 발현은 골수-단핵구성 세포(myelomonocytic cells ; 단핵구, 조직 대식세포, 쿠퍼세포(Kupffer cells), 랑게르한스세포(Langerhans cells), 소교세포(microglial cells), 용골세포(osteoclasts)들을 포함), 조혈성 전구체(hematopoietic precursors) 및 영양아층(trophoblasts)들로 제한된다. 아라이와 그의 동료들(Arai et al.)의 1999, J. Exp. Med. 190:1741-1754; 다이와 그의 동료들(Dai et al.)의 2002, Blood 99:111-120; 픽스레이와 스탠리(Pixley and Stanley)의 2004, Trends Cell Biol. 14:628-638들을 참조하시오. c-fms의 발현은 또한 일부 종양 세포들 내에서 입증되었다(기무라와 그의 동료들(Kirma et al.)의 2007, Cancer Res 67:1918-1926). 돌연변이 생쥐의 다양한 시험관 내 연구들 및 분석들에서 CSF-1이 c-fms에 대한 리간드임이 입증되었다(예를 들면, 보우레테와 로슈나이더(Bourette and Rohrschneider)의 2000, Growth Factors 17:155-166; 빅토르-예드르체흐자크와 그의 동료들(Wiktor-Jedrzejczak et al.)의 1990, Proc. Natl. Acad. Sci. U.S.A. 87:4828-4832; 요시다와 그의 동료들(Yoshida et al.)의 1990, Nature 345:442-444; 반 베센비크와 반 훌(van Wesenbeeck and van Hul)의 2005, Crit. Rev. Eukaryot. Gene Expr. 15:133-162들을 참조하시오). CSF-1의 c-fms에의 결합은 PI3-K/AKT 및 Ras/Raf/MEK/MAPK를 포함하는 신호화 경로(signaling pathways)들의 다운스트림 활성화(downstream activation)의 결과를 가져오는 특정의 사이트들에서의 상기 수용체의 자가인산화(autophosphorylation)를 유도하고 그리고 마크로파지 분화는 일차적으로 지속성 MEK 활성을 통하여 조정된다(고세와 그의 동료들(Gosse et al.)의 2005, Cellular Signaling 17:1352-1362). 인터루킨-34(IL-34)가 또한 c-fms에 대한 리간드라는 것이 매우 최근의 증거에서 나타나고 있다(린과 그의 동료들(Lin, et al.)의 2008, Science 320:807-811).
본 발명은 인간 C-FMS 항원 결합 단백질을 제공하는 것이다.
인간 c-fms를 포함하여 c-fms를 결합하는 항원-결합 단백질들이 본 명세서에서 기술된다. 상기 인간 c-fms 항원-결합 단백질들은 c-fms와 관련한 생물학적 반응들 중의 적어도 하나를 저해하거나, 간섭하거나 또는 조절한다는 것이 밝혀졌으며, 마찬가지로 c-fms-관련 질병들 또는 장애들의 영향을 완화시키는 데 유용하다. 따라서, 특정의 항원-결합 단백질들의 c-fms에의 결합은 c-fms-CSF-1 결합 또는 신호화의 저해, 간섭 또는 조절, c-fms-IL-34 결합 또는 신호화의 저해, 종양들 내로의 단핵구 이동의 감소 및/또는 종양-연관 마크로파지(TAMs)의 축적의 감소 등의 활성들 중의 하나 또는 그 이상을 가질 수 있다.
하나의 구체예에는 c-fms 수용체 항원 결합 단백질들의 생산을 위한 세포주 및 인간 c-fms에 연관된 질병들의 진단 및 치료방법들을 포함하는 발현 시스템들이 포함된다.
단리된 항원-결합 단백질들 중의 일부에는 (A) (i) SEQ ID NOs: 136-147들로 이루어지는 그룹으로부터 선택되는 CDRH1; (ii) SEQ ID NOs: 148-164들로 이루어지는 그룹으로부터 선택되는 CDRH2; (iii) SEQ ID NOs: 165-190들로 이루어지는 그룹으로부터 선택되는 CDRH3; 및 (iv) 하나 또는 그 이상의 아미노산 치환, 결손 또는 삽입들을 포함하며 집합적으로 총 4개 이하의 아미노산들인 (i), (ii) 및 (iii)의 CDRH;들로 이루어지는 그룹으로부터 선택되는 하나 또는 그 이상의 중쇄보족결정영역(heavy chain complementary determining regions ; CDRHs); (B) (i) SEQ ID NOs: 191-210들로 이루어지는 그룹으로부터 선택되는 CDRLl; (ii) SEQ ID NOs: 211-224들로 이루어지는 그룹으로부터 선택되는 CDRL2; (iii) SEQ ID NOs: 225-246들로 이루어지는 그룹으로부터 선택되는 CDRL3; 및 (iv) 하나 또는 그 이상의 아미노산 치환, 결손 또는 삽입들을 포함하며 집합적으로 총 4개 이하의 아미노산들인 (i), (ii) 및 (iii)의 CDRL;들로 이루어지는 그룹으로부터 선택되는 하나 또는 그 이상의 경쇄보족결정영역(light chain complementary determining regions ; CDRLs); 또는 (C) (A)의 하나 또는 그 이상의 중쇄 CDRHs 및 (B)의 하나 또는 그 이상의 경쇄 CDRLs들이 포함된다.
하나의 구체예에 있어서, 상기 단리된 항원-결합 단백질에는 앞서 언급한 (A)의 적어도 하나 또는 두 개의 CDRH 및 앞서 언급한 (B)의 적어도 하나 또는 두 개의 CDRL이 포함될 수 있다. 또 다른 관점에 있어서, 상기 단리된 항원-결합 단백질에는 CDRHl, CDRH2, CDRH3, CDRLl, CDRL2 및 CDRL3들이 포함된다.
특정의 항원 결합 단백질들에 있어서, 앞서 언급한 (A)의 상기 CDRH는 (i) SEQ ID NOs: 136-147들로 이루어지는 그룹으로부터 선택되는 CDRHl; (ii) SEQ ID NOs: 148-164들로 이루어지는 그룹으로부터 선택되는 CDRH2; (iii) SEQ ID NOs: 165-190들로 이루어지는 그룹으로부터 선택되는 CDRH3; 및 (iv) 2개 이하의 아미노산들의 하나 또는 그 이상의 아미노산 치환, 탈락 또는 삽입들을 포함하는 (i), (ii) 및 (iii)의 CDRH;들로 이루어지는 그룹으로부터 선택되거나; 앞서 언급한 (B)의 상기 CDRL은 (i) SEQ ID NOs: 191-210들로 이루어지는 그룹으로부터 선택되는 CDRLl; (ii) SEQ ID NOs: 211-224들로 이루어지는 그룹으로부터 선택되는 CDRL2; (iii) SEQ ID NOs: 225-246들로 이루어지는 그룹으로부터 선택되는 CDRL3 아미노산 시퀀스; 및 (iv) 2개 이하의 아미노산들의 하나 또는 그 이상의 아미노산 치환, 탈락 또는 삽입들을 포함하는 (i), (ii) 및 (iii)의 CDRL;들로 이루어지는 그룹으로부터 선택되거나; 또는 (C) (A)의 하나 또는 그 이상의 중쇄 CDRHs 및 (B)의 하나 또는 그 이상의 경쇄 CDRLs들로 이루어지는 그룹으로부터 선택된다.
또 다른 구체예에 있어서, 상기 단리된 항원-결합 단백질은 (A) (i) SEQ ID NOs: 136-147들로 이루어지는 그룹으로부터 선택되는 CDRHl; (ii) SEQ ID NOs: 148-164들로 이루어지는 그룹으로부터 선택되는 CDRH2; 및 (iii) SEQ ID NOs: 165-190들로 이루어지는 그룹으로부터 선택되는 CDRH3;들로 이루어지는 그룹으로부터 선택되는 CDRH; (B) (i) SEQ ID NOs: 191-210들로 이루어지는 그룹으로부터 선택되는 CDRLl; (ii) SEQ ID NOs: 211-224들로 이루어지는 그룹으로부터 선택되는 CDRL2; 및 (iii) SEQ ID NOs: 225-246들로 이루어지는 그룹으로부터 선택되는 CDRL3;들로 이루어지는 그룹으로부터 선택되는 CDRL; 또는 (C) (A)의 하나 또는 그 이상의 중쇄 CDRHs 및 (B)의 하나 또는 그 이상의 경쇄 CDRLs들을 포함할 수 있다. 하나의 구체예에 있어서, 상기 단리된 항원-결합 단백질에는 (A) SEQ ID NOs:136-147의 CDRHl, SEQ ID NOs: 148-164의 CDRH2, 및 SEQ ID NOs: 165-190의 CDRH3, 및 (B) SEQ ID NOs: 191-210의 CDRLl, SEQ ID NOs:211-224의 CDRL2, 및 SEQ ID NOs: 225-246의 CDRL3들이 포함될 수 있다. 또 다른 구체예에 있어서, 가변중쇄(variable heavy chain ; VH)는 SEQ ID NOs: 70-101로 이루어지는 그룹으로부터 선택되는 아미노산 시퀀스와 적어도 90%의 시퀀스 동일성을 갖거나 및/또는 가변경쇄(variable light chain ;VL)는 SEQ ID NOs: 102-135로 이루어지는 그룹으로부터 선택되는 아미노산 시퀀스와 적어도 90%의 시퀀스 동일성을 갖는다. 또 다른 구체예에 있어서, 상기 VH는 SEQ ID NOs:70-101들로 이루어지는 그룹으로부터 선택되거나 및/또는 상기 VL은 SEQ ID NOs: 102-135들로 이루어지는 그룹으로부터 선택된다.
또 다른 관점에 있어서, 인간 c-fms의 c-fms 서브도메인 Ig-형 1-1 및 Ig-형 1-2를 포함하는 에피토프(epitope)에 특이적으로 결합하는 단리된 항원 결합 단백질이 제공된다.
또 다른 관점에 있어서, (A) (i) SEQ ID NOs: 136-147에 적어도 80%의 시퀀스 동일성을 갖는 CDRHl; (ii) SEQ ID NOs: 148-164에 적어도 80%의 시퀀스 동일성을 갖는 CDRH2; 및 (iii) SEQ ID NOs: 165-190에 적어도 80%의 시퀀스 동일성을 갖는 CDRH3;들로 이루어지는 그룹으로부터 선택되는 하나 또는 그 이상의 중쇄 CDRs(CDRHs); (B) (i) SEQ ID NOs: 191-210에 적어도 80%의 시퀀스 동일성을 갖는 CDRLl; (ii) SEQ ID NOs: 211-224에 적어도 80%의 시퀀스 동일성을 갖는 CDRL2; 및 (iii) SEQ ID NOs: 225-246에 적어도 80%의 시퀀스 동일성을 갖는 CDRL3;들로 이루어지는 그룹으로부터 선택되는 하나 또는 그 이상의 경쇄 CDRs(CDRLs); 또는 (C) (A)의 하나 또는 그 이상의 중쇄 CDRHs 및 (B)의 하나 또는 그 이상의 경쇄 CDRLs들을 포함하는, c-fms를 결합하는 단리된 항원 결합 단백질이 제공된다. 하나의 구체예에 있어서, 상기 단리된 항원-결합 단백질에는 (A) (i) SEQ ID NOs: 136-147에 적어도 90%의 시퀀스 동일성을 갖는 CDRHl; (ii) SEQ ID NOs: 148-164에 적어도 90%의 시퀀스 동일성을 갖는 CDRH2; 및 (iii) SEQ ID NOs: 165-190에 적어도 90%의 시퀀스 동일성을 갖는 CDRH3;들로 이루어지는 그룹으로부터 선택되는 하나 또는 그 이상의 CDRHs; (B) (i) SEQ ID NOs: 191-210에 적어도 90%의 시퀀스 동일성을 갖는 CDRLl; (ii) SEQ ID NOs:211-224에 적어도 90%의 시퀀스 동일성을 갖는 CDRL2; 및 (iii) SEQ ID NOs: 225-246에 적어도 90%의 시퀀스 동일성을 갖는 CDRL3;들로 이루어지는 그룹으로부터 선택되는 하나 또는 그 이상의 CDRLs; 또는 (C) (A)의 하나 또는 그 이상의 중쇄 CDRHs 및 (B)의 하나 또는 그 이상의 경쇄 CDRLs들이 포함된다.
또 다른 구체예는 이하에서 기술되는 합치 시퀀스(consensus sequences)들을 갖는 CDRs들 중의 하나 또는 조합을 포함하며, c-fms와 결합하는 단리된 항원-결합 단백질이다. 그룹 A, 그룹 B 및 그룹 C는 계통발생적으로 연관된 클론(phylogenetically related clones)들로부터 유도되는 시퀀스들을 의미한다. 하나의 구체예에 있어서, 여러 그룹들로부터의 상기 CDRs들은 혼합되고 그리고 매치될 수 있다. 또 다른 관점에 있어서, 상기 항원 결합 단백질에는 하나의 그리고 동일한 그룹 A, 그룹 B 또는 그룹 C들로부터의 둘 또는 그 이상의 CDRHs들이 포함된다. 또 다른 관점에 있어서, 상기 항원 결합 단백질에는 동일한 그룹 A, 그룹 B 또는 그룹 C들로부터의 둘 또는 그 이상의 CDRLs들이 포함된다. 또 다른 관점에 있어서, 상기 항원 결합 단백질에는 동일한 그룹 A, 그룹 B 또는 그룹 C들로부터의 적어도 둘 또는 세 개의 CDRHs 및/또는 적어도 둘 또는 세 개의 CDRLs들이 포함된다. 서로 다른 그룹들에 대한 합치 시퀀스들은 다음과 같다:
그룹 A : (a) 일반식 GYTX1TSYGIS(SEQ ID NO: 307)의 CDRH1, 여기에서 X1은 F 및 L로 이루어지는 그룹으로부터 선택되는 것이고; (b) 일반식 WISAYNGNX1NYAQKX2QG(SEQ ID NO: 308)의 CDRH2, 여기에서 X1은 T 및 P로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X2는 L 및 F로 이루어지는 그룹으로부터 선택되는 것이고; (c) 일반식 X1X2X3X4X4X5FGEX6X7X8X9FDY(SEQ ID NO: 309)의 CDRH3, 여기에서 X1은 E 및 D로 이루어지는 그룹으로부터 선택되는 것이고, X2는 S 및 Q로 이루어지는 그룹으로부터 선택되는 것이고, X3은 G 및 무 아미노산(no amino acid)으로 이루어지는 그룹으로부터 선택되는 것이고, X4는 L 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X5는 W 및 G로 이루어지는 그룹으로부터 선택되는 것이고, X6은 V 및 L로 이루어지는 그룹으로부터 선택되는 것이고, X7은 E 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X8은 G 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X9는 F 및 L로 이루어지는 그룹으로부터 선택되는 것이고; (d) 일반식 KSSX1GVLX2SSX3NKNX4LA(SEQ ID NO: 310)의 CDRL1, 여기에서 X1은 Q 및 S로 이루어지는 그룹으로부터 선택되는 것이고, X2는 D 및 Y로 이루어지는 그룹으로부터 선택되는 것이고, X3은 N 및 D로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X4는 F 및 Y로 이루어지는 그룹으로부터 선택되는 것이고; (e) 일반식 WASX1RES(SEQ ID NO: 311)의 CDRL2, 여기에서 X1은 N 및 T로 이루어지는 그룹으로부터 선택되는 것이고; 그리고 (f) 일반식 QQYYX1X2PX3T(SEQ ID NO:312)의 CDRL3, 여기에서 X1은 S 및 T로 이루어지는 그룹으로부터 선택되는 것이고, X2는 D 및 T로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X3은 F 및 P로 이루어지는 그룹으로부터 선택되는 것이다.
그룹 B : (a) 일반식 GFTX1X2X3AWMS(SEQ ID NO: 313)을 갖는 CDRHl, 여기에서 X1은 F 및 V로 이루어지는 그룹으로부터 선택되는 것이고, X2는 S 및 N으로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X3은 N 및 T로 이루어지는 그룹으로부터 선택되는 것이고; (b) 일반식 RIKX1KTDGX2TX3DX4AAPVKG(SEQ ID NO: 314)을 갖는 CDRH2, 여기에서 X1은 S 및 T로 이루어지는 그룹으로부터 선택되는 것이고, X2는 G 및 W로 이루어지는 그룹으로부터 선택되는 것이고, X3은 T 및 A로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X4는 Y 및 N으로 이루어지는 그룹으로부터 선택되는 것이고; (c) 일반식 X1X2X3X4X5X6X7X8X9X10X11X12X13YYGX14DV(SEQ ID NO: 315)을 갖는 CDRH3, 여기에서 X1은 E, D 및 G로 이루어지는 그룹으로부터 선택되는 것이고, X2는 Y, L 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X3은 Y, R, G 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X4는 H, G, S 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X5는 I, A, L 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X6은 L, V, T, P 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X7은 T, V, Y, G, W 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X8은 G, V, S 및 T로 이루어지는 그룹으로부터 선택되는 것이고, X9는 S, T, D, N 및 G로 이루어지는 그룹으로부터 선택되는 것이고, X10은 G, F, P 및 Y로 이루어지는 그룹으로부터 선택되는 것이고, X11은 G, Y 및 N으로 이루어지는 그룹으로부터 선택되는 것이고, X12는 V 및 Y로 이루어지는 그룹으로부터 선택되는 것이고, X13은 W, S 및 Y로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X14는 M, T 및 V로 이루어지는 그룹으로부터 선택되는 것임; (d) 일반식 QASQDIX1NYLN(SEQ ID NO: 316)을 갖는 CDRLl, 여기에서 X1은 S 및 N으로 이루어지는 그룹으로부터 선택되는 것이고; (e) 일반식 DX1SNLEX2(SEQ ID NO: 317)을 갖는 CDRL2, 여기에서 X1은 A 및 T로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X2는 T 및 P로 이루어지는 그룹으로부터 선택되는 것이고; 그리고 (f) 일반식 QQYDX1LX2T(SEQ ID NO: 318)을 갖는 CDRL3, 여기에서 X1은 N 및 D로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X2는 L 및 I로 이루어지는 그룹으로부터 선택되는 것이다.
그룹 C : (a) 일반식 GFTFX1SYGMH(SEQ ID NO: 319)을 갖는 CDRHl, 여기에서 X1은 S 및 I로 이루어지는 그룹으로부터 선택되는 것이고; (b) 일반식 VIWYDGSNX1YYADSVKG(SEQ ID NO: 320)을 갖는 CDRH2, 여기에서 X1은 E 및 K로 이루어지는 그룹으로부터 선택되는 것이고; (c) 일반식 SSX1X2X3YX4MDV(SEQ ID NO: 321)을 갖는 CDRH3, 여기에서 X1은 G, S 및 W로 이루어지는 그룹으로부터 선택되는 것이고, X2는 N, D 및 S로 이루어지는 그룹으로부터 선택되는 것이고, X3은 Y 및 F로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X4는 D 및 G로 이루어지는 그룹으로부터 선택되는 것이고; (d) 일반식 QASX1DIX2NX3LN(SEQ ID NO: 322)을 갖는 CDRLl, 여기에서 X1은 Q 및 H로 이루어지는 그룹으로부터 선택되는 것이고, X2는 S 및 N으로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X3은 F 및 Y로 이루어지는 그룹으로부터 선택되는 것이고; (e) 일반식 DASNLEX1(SEQ ID NO: 323)을 갖는 CDRL2, 여기에서 X1은 T 및 I로 이루어지는 그룹으로부터 선택되는 것이고; 그리고 (f) 일반식 QX1YDX2X3PX4T(SEQ ID NO: 324)를 갖는 CDRL3, 여기에서 X1은 Q 및 R로 이루어지는 그룹으로부터 선택되는 것이고, X2는 N 및 D로 이루어지는 그룹으로부터 선택되는 것이고, X3은 L 및 F로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X4는 F, L 및 I로 이루어지는 그룹으로부터 선택되는 것이다.
또 다른 구체예에 있어서, 이상에서 기술된 상기 단리된 항원 결합 단백질에는 적어도 하나의 CDRH를 포함하는 제1아미노산 시퀀스 및 적어도 하나의 CDRL을 포함하는 제2아미노산 시퀀스가 포함된다. 하나의 구체예에 있어서, 상기 제1아미노산 시퀀스 및 제2아미노산 시퀀스들은 서로 공유적으로 결합된다. 또 다른 구체예에 있어서, 상기 단리된 항원-결합 단백질의 상기 제1아미노산 시퀀스에는 SEQ ID NOs: 165-190의 CDRH3, SEQ ID NOs: 148-164의 CDRH2 그리고 SEQ ID NOs: 136-147의 CDRHl들이 포함되고, 그리고 상기 단리된 항원 결합 단백질의 상기 제2아미노산 시퀀스에는 SEQ ID NOs: 225-246의 CDRL3, SEQ ID NOs: 211-224의 CDRL2 그리고 SEQ ID NOs: 191-210의 CDRLl들이 포함된다.
하나의 관점에 있어서, 본 발명에서 제공되는 상기 단리된 항원-결합 단백질은 모노클로날 항체, 폴리클로날 항체, 재조합 항체, 인간 항체, 인간화된 항체, 키메라 항체(chimeric antibody), 다중특이성 항체(multispecific antibody) 또는 이들의 항체 단편이 될 수 있다. 다른 구체예에 있어서, 상기 단리된 항원-결합 단백질들의 상기 항체 단편은 Fab 단편, Fab' 단편, F(ab')2 단편, Fv 단편, 다이어바디(diabody) 또는 단일쇄 항체 분자(single chain antibody molecule)가 될 수 있다. 또 다른 구체예에 있어서, 상기 단리된 항원 결합 단백질은 인간 항체이며, IgGl-형, IgG2-형, IgG3-형 또는 IgG4-형이 될 수 있다.
또 다른 관점에 있어서, 상기 단리된 항원-결합 단백질은 인간 c-fms의 세포외 부분(extracellular portion)에의 결합에 대하여 제공된 상기 단리된 항원-결합 단백질들 중의 하나의 항원 결합 단백질과 경쟁할 수 있다. 하나의 구체예에 있어서, 상기 단리된 항원 결합 단백질은 환자에 투여되는 경우에 단핵구 주화성(monocyte chemotaxis)을 감소시키거나, 종양들 내로의 단핵구 이동을 저해하거나, 종양 내에서의 종양 관련 마크로파지의 축적을 저해하거나 또는 질병 조직 내에서의 마크로파지의 축적을 저해할 수 있다.
또 다른 관점에 있어서, c-fms에 결합하는 상기 항원-결합 단백질들을 인코딩하는 단리된 핵산 분자들이 또한 제공된다. 일부 경우들에 있어서, 상기 단리된 핵산 분자들은 제어 시퀀스(control sequence)에 작동가능하게 연결된다.
또 다른 관점에 있어서, 앞서 언급한 c-fms에 결합하는 항원-결합 단백질들을 인코딩하는 단리된 핵산 분자들을 포함하는 발현벡터로 형질전환되거나 또는 감염된 발현벡터들 및 숙주세포들이 또한 제공된다.
또 다른 관점에 있어서, 상기 항원-결합 단백질을 분비하는 숙주세포로부터 상기 항원 결합 단백질을 준비하는 단계를 포함하는 상기 항원 결합 단백질들의 제조방법이 또한 제공된다.
또 다른 관점에 있어서, 앞서 언급한 항원-결합 단백질들 중의 적어도 하나 및 약제학적으로 수용가능한 부형제(excipient)를 포함하는 약제학적 조성물이 제공된다. 하나의 구체예에 있어서, 상기 약제학적 조성물은 방사성 동위원소(radioisotope), 방사성 핵종(radionuclide), 톡신(toxin) 또는 치료 및 화학요법군(therapeutic and a chemotherapeutic group)들로 이루어지는 그룹으로부터 선택되는 별도의 활성약품(active agent)이 포함될 수 있다.
본 발명의 구체예들에는 환자에게 유효량의 적어도 하나의 단리된 항원-결합 단백질을 투여하는 것을 포함하는, 환자에서의 c-fms 연관 상태의 치료 또는 예방을 위한 방법들이 더 제공된다. 하나의 구체예에 있어서, 상기 상태는 유방암(breast cancer), 전립선암(prostate cancer), 결장암(colorectal cancer), 자궁내막선암종(endometrial adenocarcinoma), 백혈병(leukemia), 림프종(lymphoma), 악성흑색종(melanoma), 식도의 편평상피세포암(esophageal squamous cell cancer), 위암(gastric cancer), 성상세포암(astrocytic cancer), 자궁내막암(endometrial cancer), 경부암(cervical cancer), 방광암(bladder cancer), 신장암(renal cancer), 방광암, 폐암(lung cancer) 및 난소암(ovarian cancer)들로 이루어지는 그룹으로부터 선택되는 암이다.
또 다른 관점에 있어서, 본 발명은 환자에게 유효량의 적어도 하나의 본 발명에서 제공되는 상기 항원-결합 단백질을 투여하는 것을 포함하는, 환자에서의 c-fms의 세포외 부분에의 CSF-1의 결합을 저해하는 방법이 제공된다.
또 다른 관점에 있어서, 유효량의 적어도 하나의 본 발명에서 제공되는 항원 결합 단백질의 투여를 포함하는, 환자에서의 인간 c-fms의 자가인산화를 저해하는 방법이 또한 제공된다.
또 다른 관점으로서, 유효량의 적어도 하나의 항원 결합 단백질을 투여하는 것을 포함하는, 환자에서의 단핵구 주화성을 감소시키는 방법이 더 제공된다.
하나의 관점에 있어서, 유효량의 적어도 하나의 항원 결합 단백질을 투여하는 것을 포함하는, 환자에서의 종양들 내로의 단핵구 이동을 저해하는 방법이 더 제공된다.
또 다른 관점에 있어서, 유효량의 적어도 하나의 항원 결합 단백질을 투여하는 것을 포함하는, 환자에서의 종양 내에서의 종양 연관 마크로파지의 축적을 저해하는 방법이 또한 제공된다.
이들 및 다른 관점들이 본 명세서에서 보다 상세하게 기술될 것이다. 제공된 상기 관점들 각각은 본 명세서에서 제공된 다양한 구체예들을 포함할 수 있다. 따라서 하나의 구성요소 또는 구성요소들의 조합들을 포함하는 각 구체예들이 기술된 각 관점에 포함될 수 있다는 것이 고려되어야 한다. 기술된 다른 특징들, 목적들 및 잇점들은 이하의 상세한 설명에서 명백하게 될 것이다.
본 발명은 인간 C-FMS 항원 결합 단백질을 제공하는 효과가 있다.
도 1a는 본 명세서에서 제공되는 중쇄가변영역들의 시퀀스 비교를 나타내는 도면이다. 도 1b는 본 명세서에서 제공되는 경쇄가변영역들의 시퀀스 비교를 나타내는 도면이다. CDR 및 골격영역(framework regions)들이 표시되었다.
도 2는 29개의 항-c-fms 하이브리도마(29 anti-c-fms hybridomas)들에 대한 계보분석(lineage analysis)을 나타내는 도면이다. 모든 복제된 하이브리도마들의 상기 가변중쇄(VH) 또는 가변경쇄(VL) 영역들 중의 어느 하나에 대응하는 아미노산 시퀀스들을 정렬시키고 그리고 다른 것과 비교하여 항체 다양성을 분석하였다. 이들 비교 정렬들을 나타내는 계통수(dendrograms)들을 나타내었으며, 여기에서 수평 가지 길이는 임의의 두 시퀀스들 또는 시퀀스 분기군들(sequence clades ; 밀접하게 ㅇr관된 시퀀스들의 그룹) 사이의 치환들(차이점들)의 상대적인 수에 대응한다. 합치 시퀀스들의 결정을 위하여 함께 그룹화된 시퀀스들이 나타나 있다.
도 3은 여러 하이브리도마 항-c-fms 상청액(supernatants)들에 의한 AML-5 증식의 저해를 입증하는 도면이다. 도 3a는 하이브리도마 항-c-fms 상청액으로의 AML-5 생물검정(bioassary)을 나타내는 도면이다. 도 3b는 정제된 재조합 항-c-fms 항체들로의 AML-5 생물검정(bioassay)을 나타내는 도면이다. AML-5 세포들은 감소되는 농도들의 항체의 존재 중에서 10ng/㎖ CSF-1으로 배양시켰다. 72시간 후, 세포 증식을 알라마르 블루(Alamar Blue)를 사용하여 측정하였다.
도 4는 CSF-1 내에서의 c-fms 항체들의 적정으로의 사이노비엠 분석(CynoBM assay)을 나타내는 도면이다. 여러 하이브리도마 항-c-fms 상청액들에 의한 CSF-1 풍부 사이노몰거스(cynomolgus ; 필리핀 원숭이) 골수세포 증식의 저해가 설명되어 있다. 사이노몰거스 골수세포들은 감소되는 농도들의 항체의 존재 중에서 10ng/㎖ CSF-1으로 배양시켰다. 72시간 후, 세포 증식을 알라마르 블루를 사용하여 측정하였다.
도 5는 IgG2 mAbs(PT, 부모형들(parent forms))에 의한 리간드-유도 pTyr-c-fms의 저해를 나타내는 도면이다. 293T/c-fms 세포들을 1시간 동안 혈청-결핍시키고, 그리고 IgG2 mAbs, 1.109, 1.2 또는 2.360(PT) 및 대조 mAbs 항-c-fms 3-4A4(비-차단) 및 항-h-CD39 M 105(비-특이적)를 일련의 적정(1.0 내지 0.0001㎍/㎖) 또는 1.0㎍/㎖(대조)로 처리하였다. 계속해서 세포들을 50ng/㎖ CSF-1로 5분 동안 37℃에서 자극시켰다. 전체 세포 용해물(lysates)들을 기술된 바와 같이 항-c-fms C20으로 면역침강(immunoprecipitated) 시켰다. 웨스턴블럿(western blots)으로 pTyr/c-fms 및 총 c-fms 각각의 검출을 위한 항-pTyr 4G10(상부 패널) 또는 항-c-fms C20(하부 패널)들 중 하나에 대해 조사하였다.
도 6은 IgG2 mAbs (PT 대 SM(체세포 돌연변이 큐어링된 ; somatic mutation cured) 형태들)에 의한 리간드-유도 pTyr-c-fms의 저해를 비교하는 도면이다. 293T/c-fms 세포들을 1시간 동안 혈청-결핍시키고, 그리고 IgG2 mAbs, 1.109, 1.2 또는 2.360(둘 다 PT 또는 SM) 및 대조 mAbs 항-c-fms 3-4A4(비-차단)을 1.0 및 0.1㎍/㎖로 처리하였다. 계속해서 세포들을 50ng/㎖ CSF-1로 5분 동안 37℃에서 자극시키고, 전체 세포 용해물들을 기술된 바와 같이 항-c-fms C20으로 면역침강 시켰다. 웨스턴블럿으로 pTyr/c-fms 및 총 c-fms 각각의 검출을 위한 항-pTyr 4G10(상부 패널) 또는 항-c-fms C20(하부 패널)들 중 하나에 대해 조사하였다.
도 7은 IgG2 mAbs(PT 대 SM 형태들)에 의한 c-fms의 면역침강의 웨스턴블럿을 나타내는 도면이다. 미자극의 293T/c-fms 세포들의 전체 세포 용해물들을 IgG2 mAbs, 1.109, 1.2 또는 2.360(둘 다 PT 또는 SM 형태들) 및 항-c-fms C20 2.5㎍/㎖에서의 항-c-fms C20을 사용하여 4℃에서 밤새도록 면역침강시켰다. 웨스턴블럿으로 항-c-fms C20 및 항-토끼 IgG/HRP에 대해 조사하였다.
도 8은 인간 c-fms의 세포외 도메인 영역의 아미노 시퀀스(SEQ ID NO: 1)를나타내는 도면이다.
도 9는 c-fms SNPs의 면역침강의 웨스턴블럿을 나타내는 도면이다. 표시된 c-fms SNPs의 발현 구축물(expression constructs)들은 293T/c-fms 세포들 내에서 구축되고 그리고 일시적으로 발현되었다. 계속해서 미자극의 전체 세표 용해물들을 각 mAb 및 대조 Abs로 면역침강시켰다. 웨스턴블럿으로 c-fms H300 및 항-토끼에 대해 조사하였다.
도 10은 인간 c-fms ECD(세포외 도메인(extracellular domain)) 및 끝을 잘라버린 구축물(truncated constructs)들의 다이아그램을 나타내는 도면이다. c-fms의 N 말단에서 프레임 내에 아비딘 태그(avidin tag)가 융합되었다. 각 c-fms 구축물들에 대하여 처음과 마지막 4개의 아미노산들이 표시되었다.
도 11은 FITC 표지 항-아비딘, 1.109, 1.2 및 2.360 c-fms 항체들의 c-fms ECD 및 끝이 절단된 아비딘 융합 단백질에의 결합을 입증하는 도면이다.
도 12는 항-아비딘 FITC, 대조 항체 및 항-c-fms 항체들(FITC 표지된)의 전장(full length)의 c-fms 및 Ig-형 루프2(단독) 융합 단백질에의 결합을 나타내는 도면이다.
도 13은 20배의 미표지된 1.109, 1.2 및 2.360 c-fms 항체들 및 후속하여 1㎍/㎖ 농도의 FITC 표지된 1.109에 대한 경쟁 분석(competition assay)을 나타내는 도면이다.
도 14는 20배의 미표지된 1.109, 1.2 및 2.360 c-fms 항체들 및 후속하여 1㎍/㎖ 농도의 FITC 표지된 1.2에 대한 경쟁 분석을 나타내는 도면이다.
도 15는 20배의 미표지된 1.109, 1.2 및 2.360 c-fms 항체들 및 후속하여 1㎍/㎖ 농도의 FITC 표지된 2.360에 대한 경쟁 분석을 나타내는 도면이다.
도 16은 각 종양의 종양 용적 및 괴사율(percent necrosis)을 측정하는 수단으로서의 항-쥣과 동물 c-fms 항체(anti-murine c-fms antibody)에 의한 MDAMB231 유방선암(breast adenocarcinoma) 이종이식의 성장의 저해를 나타내는 도면이다. 계속해서 각 종양의 괴사율을 이들 측정치들로부터 계산하였으며, 도 16에 나타내었다.
도 17은 구축된 NCIHl 975 폐선암 이종이식의 성장의 저해를 나타내는 도면이다. 항-쥣과 동물 c-fms 항체가 구축된 NCIH 1975 폐선암 이종이식의 성장을 저해할 수 있다는 것을 입증하는 종양 측정들 및 치료일수들이 나타나 있다.
도 2는 29개의 항-c-fms 하이브리도마(29 anti-c-fms hybridomas)들에 대한 계보분석(lineage analysis)을 나타내는 도면이다. 모든 복제된 하이브리도마들의 상기 가변중쇄(VH) 또는 가변경쇄(VL) 영역들 중의 어느 하나에 대응하는 아미노산 시퀀스들을 정렬시키고 그리고 다른 것과 비교하여 항체 다양성을 분석하였다. 이들 비교 정렬들을 나타내는 계통수(dendrograms)들을 나타내었으며, 여기에서 수평 가지 길이는 임의의 두 시퀀스들 또는 시퀀스 분기군들(sequence clades ; 밀접하게 ㅇr관된 시퀀스들의 그룹) 사이의 치환들(차이점들)의 상대적인 수에 대응한다. 합치 시퀀스들의 결정을 위하여 함께 그룹화된 시퀀스들이 나타나 있다.
도 3은 여러 하이브리도마 항-c-fms 상청액(supernatants)들에 의한 AML-5 증식의 저해를 입증하는 도면이다. 도 3a는 하이브리도마 항-c-fms 상청액으로의 AML-5 생물검정(bioassary)을 나타내는 도면이다. 도 3b는 정제된 재조합 항-c-fms 항체들로의 AML-5 생물검정(bioassay)을 나타내는 도면이다. AML-5 세포들은 감소되는 농도들의 항체의 존재 중에서 10ng/㎖ CSF-1으로 배양시켰다. 72시간 후, 세포 증식을 알라마르 블루(Alamar Blue)를 사용하여 측정하였다.
도 4는 CSF-1 내에서의 c-fms 항체들의 적정으로의 사이노비엠 분석(CynoBM assay)을 나타내는 도면이다. 여러 하이브리도마 항-c-fms 상청액들에 의한 CSF-1 풍부 사이노몰거스(cynomolgus ; 필리핀 원숭이) 골수세포 증식의 저해가 설명되어 있다. 사이노몰거스 골수세포들은 감소되는 농도들의 항체의 존재 중에서 10ng/㎖ CSF-1으로 배양시켰다. 72시간 후, 세포 증식을 알라마르 블루를 사용하여 측정하였다.
도 5는 IgG2 mAbs(PT, 부모형들(parent forms))에 의한 리간드-유도 pTyr-c-fms의 저해를 나타내는 도면이다. 293T/c-fms 세포들을 1시간 동안 혈청-결핍시키고, 그리고 IgG2 mAbs, 1.109, 1.2 또는 2.360(PT) 및 대조 mAbs 항-c-fms 3-4A4(비-차단) 및 항-h-CD39 M 105(비-특이적)를 일련의 적정(1.0 내지 0.0001㎍/㎖) 또는 1.0㎍/㎖(대조)로 처리하였다. 계속해서 세포들을 50ng/㎖ CSF-1로 5분 동안 37℃에서 자극시켰다. 전체 세포 용해물(lysates)들을 기술된 바와 같이 항-c-fms C20으로 면역침강(immunoprecipitated) 시켰다. 웨스턴블럿(western blots)으로 pTyr/c-fms 및 총 c-fms 각각의 검출을 위한 항-pTyr 4G10(상부 패널) 또는 항-c-fms C20(하부 패널)들 중 하나에 대해 조사하였다.
도 6은 IgG2 mAbs (PT 대 SM(체세포 돌연변이 큐어링된 ; somatic mutation cured) 형태들)에 의한 리간드-유도 pTyr-c-fms의 저해를 비교하는 도면이다. 293T/c-fms 세포들을 1시간 동안 혈청-결핍시키고, 그리고 IgG2 mAbs, 1.109, 1.2 또는 2.360(둘 다 PT 또는 SM) 및 대조 mAbs 항-c-fms 3-4A4(비-차단)을 1.0 및 0.1㎍/㎖로 처리하였다. 계속해서 세포들을 50ng/㎖ CSF-1로 5분 동안 37℃에서 자극시키고, 전체 세포 용해물들을 기술된 바와 같이 항-c-fms C20으로 면역침강 시켰다. 웨스턴블럿으로 pTyr/c-fms 및 총 c-fms 각각의 검출을 위한 항-pTyr 4G10(상부 패널) 또는 항-c-fms C20(하부 패널)들 중 하나에 대해 조사하였다.
도 7은 IgG2 mAbs(PT 대 SM 형태들)에 의한 c-fms의 면역침강의 웨스턴블럿을 나타내는 도면이다. 미자극의 293T/c-fms 세포들의 전체 세포 용해물들을 IgG2 mAbs, 1.109, 1.2 또는 2.360(둘 다 PT 또는 SM 형태들) 및 항-c-fms C20 2.5㎍/㎖에서의 항-c-fms C20을 사용하여 4℃에서 밤새도록 면역침강시켰다. 웨스턴블럿으로 항-c-fms C20 및 항-토끼 IgG/HRP에 대해 조사하였다.
도 8은 인간 c-fms의 세포외 도메인 영역의 아미노 시퀀스(SEQ ID NO: 1)를나타내는 도면이다.
도 9는 c-fms SNPs의 면역침강의 웨스턴블럿을 나타내는 도면이다. 표시된 c-fms SNPs의 발현 구축물(expression constructs)들은 293T/c-fms 세포들 내에서 구축되고 그리고 일시적으로 발현되었다. 계속해서 미자극의 전체 세표 용해물들을 각 mAb 및 대조 Abs로 면역침강시켰다. 웨스턴블럿으로 c-fms H300 및 항-토끼에 대해 조사하였다.
도 10은 인간 c-fms ECD(세포외 도메인(extracellular domain)) 및 끝을 잘라버린 구축물(truncated constructs)들의 다이아그램을 나타내는 도면이다. c-fms의 N 말단에서 프레임 내에 아비딘 태그(avidin tag)가 융합되었다. 각 c-fms 구축물들에 대하여 처음과 마지막 4개의 아미노산들이 표시되었다.
도 11은 FITC 표지 항-아비딘, 1.109, 1.2 및 2.360 c-fms 항체들의 c-fms ECD 및 끝이 절단된 아비딘 융합 단백질에의 결합을 입증하는 도면이다.
도 12는 항-아비딘 FITC, 대조 항체 및 항-c-fms 항체들(FITC 표지된)의 전장(full length)의 c-fms 및 Ig-형 루프2(단독) 융합 단백질에의 결합을 나타내는 도면이다.
도 13은 20배의 미표지된 1.109, 1.2 및 2.360 c-fms 항체들 및 후속하여 1㎍/㎖ 농도의 FITC 표지된 1.109에 대한 경쟁 분석(competition assay)을 나타내는 도면이다.
도 14는 20배의 미표지된 1.109, 1.2 및 2.360 c-fms 항체들 및 후속하여 1㎍/㎖ 농도의 FITC 표지된 1.2에 대한 경쟁 분석을 나타내는 도면이다.
도 15는 20배의 미표지된 1.109, 1.2 및 2.360 c-fms 항체들 및 후속하여 1㎍/㎖ 농도의 FITC 표지된 2.360에 대한 경쟁 분석을 나타내는 도면이다.
도 16은 각 종양의 종양 용적 및 괴사율(percent necrosis)을 측정하는 수단으로서의 항-쥣과 동물 c-fms 항체(anti-murine c-fms antibody)에 의한 MDAMB231 유방선암(breast adenocarcinoma) 이종이식의 성장의 저해를 나타내는 도면이다. 계속해서 각 종양의 괴사율을 이들 측정치들로부터 계산하였으며, 도 16에 나타내었다.
도 17은 구축된 NCIHl 975 폐선암 이종이식의 성장의 저해를 나타내는 도면이다. 항-쥣과 동물 c-fms 항체가 구축된 NCIH 1975 폐선암 이종이식의 성장을 저해할 수 있다는 것을 입증하는 종양 측정들 및 치료일수들이 나타나 있다.
본 명세서에서 사용되는 절의 두문(section headings)들은 단지 조직적인 목적들을 위한 것이며, 기술된 대상 사안을 제한하는 것으로 해석되어서는 안될 것이다.
본 명세서에서 달리 정의하지 않는 한, 본 출원과 관련하여 사용된 과학적 및 기술적인 용어들은 당해 기술분야에서 통상의 지식을 가진 자에게 통상적으로 이해될 수 있는 것이다. 더욱이, 문맥(context)에 의해 달리 요구되지 않는 한, 단수단어들은 복수를 포함하며, 복수단어들은 단수를 포함할 수 있다.
일반적으로, 세포 및 조직 배양, 분자생물학, 면역학, 미생물학, 유전학 및 단백질과 핵산 화학들과 관련하여 사용된 명명법 및 기술들 그리고 본 명세서에서 기술된 잡종형성(hybridization)들은 당해 기술분야에서 공지된 것이며 또한 통상적으로 사용되는 것들이다. 본 출원의 방법들 및 기술들은 달리 표시되지 않은 한, 대체로 당해 기술분야에서 공지되고 그리고 여러 일반적인 본 명세서를 통하여 언급되고 그리고 기술된 보다 특이한 참조문헌들에 따라 수행된다. 예를 들면, 본 명세서에서 참조로 인용되는 샘브룩과 그의 동료들(Sambrook et al)의 Molecular Cloning: A Laboratory Manual, 3rd ed., Cold Spring Harbor Laboratory Press, Cold Spring Harbor, N. Y. (2001), 오우수벨과 그의 동료들(Ausubel et al)의 Current Protocols in Molecular Biology, Greene Publishing Associates (1992) 및 하로우와 레인(Harlow and Lane)의 Antibodies: A Laboratory Manual Cold Spring Harbor Laboratory Press, Cold Spring Harbor, N. Y. (1990)들을 참조하시오.
효소반응들 및 정제기술들은 당해 기술분야에서 통상적으로 수행되거나 또는 본 명세서에서 기술되는 바와 마찬가지로 제조업자들의 사용들에 따라 수행되었다. 본 명세서에서 기술된 분석화학, 합성유기화학 및 약리화학과 제약화학들과 관련되어 사용된 용어 및 실험적인 절차들과 기술들은 당해 기술분야에서 공지되고 그리고 통상적으로 사용되는 것들이다. 화학적 합성, 화학적 분석, 제약학적 제조, 제형 및 공급 및 환자들의 치료에 대해서는 표준의 기술들이 사용될 수 있다.
본 발명은 본 명세서에서 기술된 특정의 방법론, 프로토콜(protocols) 및 시약 등등에 제한되는 것이 아니며, 그것 나름으로 다양할 수 있는 것이라는 점은 이해되어야 한다. 본 명세서에서 사용된 용어는 단지 특정의 구체예들을 설명하기 위한 목적이며, 기술된 관점으로 제한되는 것이 아니며, 이는 전적으로 특허청구범위들로 한정되어야 한다.
실시하는 구체예들 이외에, 또는 달리 표시하지 않는 한, 본 명세서에서 사용된 성분들 또는 반응조건들을 나타내는 모든 숫자들은 모든 경우들에서 용어 "약"으로 변형되는 것으로 이해되어야만 한다. 상기 용어 "약"은 백분율과 관련하여 사용되는 경우 평균±1%가 될 수 있다.
정의
용어 "폴리뉴클레오티드" 또는 "핵산"은 단일-가닥 및 이중-가닥의 뉴클레오티드 중합체들 둘 다를 포함한다. 상기 폴리뉴클레오티드를 포함하는 상기 뉴클레오티드들은 리보뉴클레오티드 또는 디옥시리보뉴클레오티드 또는 뉴클레오티드의 이들 두 형태의 변형된 형태가 될 수 있다. 상기 변형들에는 브로모우리딘(bromouridine) 및 이노신 유도체들(inosine derivatives) 등과 같은 염기 변형들, 2',3'-디디옥시리보스(2',3'-dideoxyribose) 등과 같은 리보스 변형들 및 포스포로티오에이트(phosphorothioate), 포스포로디티오에이트(phosphorodithioate), 포스포로셀레노에이트(phosphoroselenoate), 포스포로디셀레노에이트(phosphorodiselenoate), 포스포로아닐로티오에이트(phosphoroanilothioate), 포스포르아닐라데이트(phoshoraniladate) 및 포스포로아미데이트(phosphoroamidate) 등과 같은 뉴클레오티드간 연결 변형들(internucleotide linkage modifications)이 포함된다.
용어 "올리고뉴클레오티드"는 200 또는 그 이하의 뉴클레오티드들을 포함하는 폴리뉴클레오티드를 의미한다. 일부 구체예들에 있어서, 올리고뉴클레오티드들은 길이에 있어서 10 내지 60개의 염기들이다. 다른 구체예들에 있어서, 올리고뉴클레오티드들은 길이에 있어서 12, 13, 14, 15, 16, 17, 18, 19 또는 20 내지 40개의 뉴클레오티드들이다. 올리고뉴클레오티드들은 예를 들면 돌연변이 유전자의 구축에서 사용하기 위한 단일 가닥 또는 이중 가닥이 될 수 있다. 올리고뉴클레오티드들은 감작(sense) 또는 비감작의 올리고뉴클레오티들이 될 수 있다. 올리고뉴클레오티드는 분석시험을 위하여 방사성표지(radiolabel), 형광표지(fluorescent label), 부착소(hapten) 또는 항원성 표지(antigenic label)를 포함하는 표지(label)를 포함할 수 있다. 예를 들면, 올리고뉴클레오티드들은 PCR 프라이머(PCR primers), 복제 프라이머(cloning primers) 또는 혼성화 프로브(hybridization probes) 들로서 사용될 수 있다.
"단리된 핵산 분자"는 게놈의 DNA 또는 RNA, mRNA, cDNA 또는 합성 유래(synthetic origin) 또는 단리된 단리된 폴리뉴클레오티드가 자연에서 발견되는 것인 폴리뉴클레오티드의 전부 또는 일부와 연관되지 않거나 또는 자연에서는 연결되지 않는 폴리뉴클레오티드에 연결되는 폴리뉴클레오티드들의 일부의 조합들을 의미한다. 이 설명을 위한 목적으로, 특정의 뉴클레오티드 시퀀스를 "포함하는 핵산 분자"는 손상되지 않은 크로모좀(intact chromosomes)들을 포함하지 않는다. 특정의 핵산 시퀀스를 "포함하는" 단리된 핵산 분자들에는 특정의 시퀀스들에 더해, 10개 이상 심지어 20개 이상의 다른 단백질들 또는 그들의 일부에 대한 코딩 시퀀스(coding sequence)가 포함될 수 있으며, 또는 언급된 핵산 시퀀스들의 코딩 영역(coding region)의 발현을 제어하는 작동가능하게 연결되는 조절 시퀀스(regulatory sequences)가 포함될 수 있거나 및/또는 벡터 시퀀스가 포함될 수 있다.
달리 특정하지 않는 한, 본 명세서에서 논의되는 임의의 단일 가닥의 폴리뉴클레오티드 시퀀스의 좌수 단부(left-hand end)는 5' 단부이고; 이중-가닥의 폴리뉴클레오티드 시퀀스들의 좌수 단부는 5' 방향들을 의미한다. 초기의 RNA 전사체들의 5'에서 3'로의 부가의 방향의 전사 방향을 의미하고; 상기 RNA 전사체의 5'에서 5' 단부인 상기 RNA 전사체와 동일한 시퀀스를 갖는 DNA 가닥 상의 시퀀스 영역은 "업스트림 시퀀스(upstream sequences)"를 의미하고; 상기 RNA 전사체의 3'에서 3' 단부인 상기 RNA 전사체와 동일한 시퀀스를 갖는 DNA 가닥 상의 시퀀스 영역은 "다운스트림 시퀀스(downstream sequences)"를 의미한다.
용어 "제어 시퀀스"는 그것이 결찰되는 코딩 시퀀스들의 발현 및 진행에 영향을 줄 수 있는 폴리뉴클레오티드 시퀀스를 의미한다. 이러한 제어 시퀀스들의 속성은 숙주 유기체에 의존적일 수 있다. 특정의 구체예들에 있어서, 원핵생물들에 대한 제어 시퀀스들에는 프로모터, 리보좀 결합 사이트 및 전사 종결 시퀀스들이 포함될 수 있다. 예를 들면, 진핵생물들에 대한 제어 시퀀스들에는 전사 인자들을 위한 하나 또는 다수의 인식 사이트들을 포함하는 프로모터, 전사 강화 시퀀스(transcription enhancer sequences) 및 전사 종결 시퀀스들이 포함될 수 있다. "제어 시퀀스"는 선도 시퀀스들(leader sequences) 및/또는 융합 짝 시퀀스(fusion partner sequences)들이 포함될 수 있다.
용어 "벡터"는 단백질 코딩 정보를 숙주세포에로 전달하는 데 사용되는 임의의 분자 또는 독립체(entity)(예를 들면, 핵산, 플라스미드, 박테리오파지 또는 바이러스)를 의미한다.
용어 "발현 벡터" 또는 "발현 구축물"은 숙주세포의 형질전환에 적절하고 그리고 그에 작동가능하게 연결되는 하나 또는 그 이상의 이종 기원의 코딩 영역의 발현을 지향하거나 및/또는 제어(숙주세포와 함께)하는 핵산 시퀀스들을 포함하는 벡터를 의미한다. 발현 구축물에는 전사, 번역에 영향을 주거나 또는 제어하고, 그리고 인트론들이 존재하는 경우 그에 작동가능하게 연결되는 코딩 영역의 RNA 접합(RNA splicing)에 영향을 주는 시퀀스들이 포함될 수 있으나, 이들에 제한되는 것은 아니다.
본 명세서에서 사용되는 바와 같이, "작동가능하게 연결되는"은 상기 용어가 적용되는 구성성분들이 적절한 조건들 하에서 이들이 이들의 고유의 기능들을 수행하는 것을 허용하는 관계에 있다는 것을 의미한다. 예를 들면, 단백질 코딩 시퀀스에 "작동가능하게 연결되는" 벡터 내의 제어 시퀀스는 그에 결찰되어 상기 제어 시퀀스들의 전사적인 활성에 적합한 조건들 하에서 상기 단백질 코딩 시퀀스의 발현이 달성되도록 한다.
용어 "숙주세포"는 핵산 시퀀스로 형질전환되거나 또는 형질전환될 수 있으며 그에 의하여 대상 유전자를 발현하는 세포를 의미한다. 상기 용어는 흥미의 대상이 되는 유전자가 존재하는 한에서는 자손(progeny)이 형태학 또는 유전적 구성(genetic make-up)에 있어서 원래의 부모세포와 동일하거나 또는 동일하지 않거나 간에 부모세포의 자손을 포함한다.
용어 "형질도입(transduction)"은 대개 박테리오파지에 의한 하나의 박테리아로부터 다른 박테리아로의 유전자들의 전달을 의미한다. "형질도입"은 또한 복제-결핍 레트로바이러스(replication defective retroviruses)들에 의한 진핵동물의 세포상 시퀀스들의 획득 및 전달을 의미한다.
용어 "형질변환(transfection)"은 세포에 의한 외래 또는 외인성의 DNA의 섭취를 의미하며, 상기 외인성의 DNA가 상기 세포막 내로 도입되는 경우에 세포는 "형질변환되었다"고 한다. 다수의 형질변환 기술들이 당해 기술분야에서 공지되어 있으며, 본 명세서에서 기술된다. 예를 들면, 그레험과 그의 동료들(Graham et al)의 1973, Virology 52:456; 샘브룩과 그의 동료들(Sambrook et al)의 2001, Molecular Cloning: A Laboratory Manual, supra; 데이비스와 그의 동료들(Davis et al)의 1986, Basic Methods in Molecular Biology, Elsevier; 추와 그의 동료들(Chu et al.)의 1981, Gene 13:197들을 참조하시오. 이러한 기술들은 하나 또는 그 이상의 외인성의 DNA 부분들을 적절한 숙주세포들 내로 도입하는 데 사용될 수 있다.
용어 "형질전환"은 세포의 유전적 특성들에서의 변화를 의미하며, 세포가 새로운 DNA 또는 RNA를 포함하도록 변형된 경우에는 세포가 형질전환되었다고 한다. 예를 들면, 형질변환, 형질도입 또는 다른 기술들을 통한 새로운 유전적 물질의 도입에 의하여 세포가 그의 원래의 상태로부터 유전적으로 변형되는 경우, 세포는 형질전환되었다고 한다. 형질변환 또는 형질도입에 후속하여, 형질전환되는 DNA는 세포의 크로모좀 내로의 물리적인 통합에 의하여 세포의 DNA와 재조합되거나, 또는 일시적으로 복제됨이 없이 에피좀의 요소(episomal element ; 유전자 부체의 요소)로서 일시적으로 유지되거나, 또는 독립적으로 플라스미드로서 복제될 수 있다. 상기 형질전환되는 DNA가 세포들의 분열과 함께 복제되는 경우 세포는 "안정하게 형질전환되었다"고 고려된다.
용어 "폴리펩티드" 또는 "단백질"은 본 명세서에서는 아미노산 잔기들의 중합체를 의미하는 것으로 상호교환적으로 사용된다. 상기 용어들은 또한 자연적으로 발생하는 아미노산 중합체들과 마찬가지로 하나 또는 그 이상의 아미노산 잔기들이 대응하는 자연적으로 발생하는 아미노산의 유사체 또는 모방체(mimetic)인 아미노산 중합체들에도 적용된다. 상기 용어들은 또한 예를 들면 탄수화물 잔기들의 도입에 의하여 당단백질을 형성하거나 또는 인산화로 변형된 아미노산 중합체들을 포함할 수 있다. 폴리펩티드들 및 단백질들은 자연적으로 발생하는 그리고 재조합 되지 않은 세포에 의해 생상될 수 있거나; 또는 유전조작되거나 또는 재조합의 세포에 의해 생산되고, 그리고 자연의 시퀀스의 하나 또는 그 이상의 아미노산들로부터의 탈락, 상기 아미노산들에의 첨가 및/또는 치환들을 갖는 자연의 단백질 또는 분자들의 아미노산 시퀀스를 갖는 분자들을 포함한다. 상기 용어 "폴리펩티드" 및 "단백질"은 특히 항원-결합 단백질의 하나 또는 그 이상의 아미노산들로부터의 탈락, 상기 아미노산들에의 첨가 및/또는 치환들을 갖는 c-fms 항원-결합 단백질들, 항체들 또는 시퀀스들을 포함한다. 용어 "폴리펩티드 단편"은 전장의 단백질(full-length protein)과 비교하여 아미노-말단 탈락, 카르복실-말단 탈락 및/또는 내부 탈락을 갖는 폴리펩티드를 의미한다. 이러한 단편들은 또한 상기 전장의 단백질과 비교하여 변형된 아미노산들을 포함할 수 있다. 특정의 구체예들에 있어서, 단편들은 대략 5 내지 500개의 아미노산 길이이다. 예를 들면, 단편들은 적어도 5, 6, 8, 10, 14, 20, 50, 70, 100, 110, 150, 200, 250, 300, 350, 400, 또는 450개의 아미노산 길이가 될 수 있다. 유용한 폴리펩티드 단편들에는 바인딩 도메인(binding domains)들을 포함하여 항체들의 면역학적으로 기능적인 단편들이 포함된다. c-fms-바인딩 항체의 경우에 있어서, 유용한 단편들에는 CDR 영역, 중쇄 및 경쇄의 가변 도메인, 항체쇄(antibody chain)의 일부 또는 2개의 CDR들을 포함하는 바로 그의 가변영역 등이 포함되나, 이들에 제한되는 것은 아니다.
용어 "단리된 단백질"은 (1) 적어도 일부의 다른 단백질들이 없고 그것으로 자연적으로 발견되거나, (2) 예를 들면, 동일한 원천, 동일한 종들로부터의 다른 단백질들이 필수적으로 없거나, (3) 서로 다른 종들로부터의 세포에 의해 발현되거나, (4) 폴리뉴클레오티드들, 지질들, 탄수화물들 또는 다른 물질들의 적어도 약 50%로부터 분리되고 그것으로 속성에 있어서 연관되거나, (5) (공유적 또는 비공유적 상호작용에 의하여) 폴리펩티드와 작동가능하게 연관되고 그것으로 자여넹서는 연관되지 않거나, 또는 (6) 자연에서는 발생되지 않는 대상체 단백질을 의미한다. 전형적으로, "단리된 단백질"은 주어진 샘플의 적어도 약 5%, 적어도 약 10%, 적어도 약 25%, 또는 적어도 약 50%를 구성한다. 합성 유래의 게놈의 DNA, cDNA, mRNA 또는 다른 RNA 또는 이들의 임의의 조합이 이러한 단리된 단백질을 인코딩할 수 있다. 바람직하게는, 상기 단리된 단백질은 그의 자연적 환경에서 발견되며, 치료적, 진단적, 예방적, 연구적 또는 다른 용도들에 대하여 방해할 수 있는 단백질들 또는 폴리펩티드들 또는 다른 오염물들이 대체로 없는 것이다.
폴리펩티드의 "변종(variant)"(예를 들면, 항원 결합 단백질 또는 항체)에는 다른 폴리펩티드 시퀀스에 비하여 하나 또는 그 이상의 아미노산 잔기들이 상기 아미노산 시퀀스들 내로 삽입되거나, 상기 시퀀스들로부터 탈락되거나 및/또는 치환되는 아미노산 시퀀스가 포함된다. 변종들에는 융합 단백질들이 포함된다.
폴리펩티드의 "유도체"는 삽입, 탈락 또는 치환 변종들과는 구별되는 몇몇 방법으로 예를 들면 다른 화학적 부분에로의 공액화(conjugation)를 통하여 화학적으로 변형된 폴리펩티드(예를 들면, 항원 결합 단백질 또는 항체)이다.
폴리펩티드, 핵산, 숙주세포 등과 같은 생물학적 물질들과 관련하여 본 명세서를 통하여 사용된 바와 같은 용어 "자연적으로 발생하는"은 자연에서 발견되는 물질들을 의미한다.
본 명세서에서 사용된 바와 같은 "항원 결합 단백질"은 c-fms 또는 인간 c-fms 등과 같은 특정의 목표 항원에 특이적으로 결합하는 단백질을 의미한다.
해리상수(dissociation constant ; KD)가 10-8 이하(≤10-8)인 경우에 항원 결합 단백질은 그의 목표 항원을 "특이적으로 결합한다"라고 한다. KD가 5*10-9M인 경우에 "높은 친화도"로, 그리고 KD가 5*10-10M인 경우에 "매우 높은 친화도"로 상기 항체는 항원에 특이적으로 결합한다. 하나의 구체예에 있어서, 상기 항체는 ≤10-9의 KD와, 대략 1*10-4/초의 오프-레이트(off-rate)를 갖는다. 하나의 구체예에 있어서, 상기 오프-레이트는 대략 1*10-5/초이다. 다른 구체예에 있어서, 상기 항체들은 대략 10-8M 내지 10-10M 사이의 KD로 c-fms 또는 인간 c-fms에 결합할 수 있으며, 또 다른 구체예에 있어서는, 이는 KD≤2*10-10으로 결합할 수 있다.
"항원 결합 영역"은 특정의 항원을 특이적으로 결합하는 단백질 또는 단백질의 일부를 의미한다. 예를 들면, 항원과 상호작용하고 그리고 상기 항원 결합 단백질에 상기 항원에 대한 그의 특이성 및 친화도를 부여하는 아미노산 잔기들을 포함하는 항원 결합 단백질의 일부를 "항원 결합 영역"으로 칭한다. 항원 결합 영역은 전형적으로 하나 또는 그 이상의 "상보적 결합 영역들(complementary binding regions" ; "CDRs")을 포함한다. 특정의 항원 결합 영역들에는 하나 또는 그 이상의 "골격(framework)" 영역들이 포함된다. "CDR"은 항원 결합 특이성 및 친화도에 기여하는 아미노산 시퀀스이다. "골격" 영역들은 상기 CDRs의 적절한 형태를 유지하는 데 도움을 주어 상기 항원 결합 영역과 항원 사이의 결합을 촉진하도록 할 수 있다.
특정의 관점들에 있어서, c-fms 단백질 또는 인간 c-fms를 결합하는 재조합 항원 결합 단백질들이 제공된다. 이러한 정황에서, "재조합 단백질"은 재조합 기술들 즉, 본 명세서에서 기술된 바와 같은 재조합 핵산의 발현을 통하여 만들어진 단백질이다. 재조합 단백질들의 생산을 위한 방법들 및 기술들은 당해 기술분야에서 공지된 것들이다.
용어 "항체"는 상기 목표 항원에의 특이적인 결합을 위한 손상되지 않은 항체와 경쟁할 수 있는 임의의 아이소타입(isotype)의 손상되지 않은 면역글로블린 또는 그의 단편을 의미하며, 예를 들면, 키메라의, 인간화된, 완전한 인간의, 그리고 양특이성 항체(bispecific antibodies)들이 포함된다. 이와 같은 "항체"는 항원 결합 단백질의 종들이다. 손상되지 않은 항체는 대체로 적어도 2개의 전장의 중쇄들 및 2개의 전장의 경쇄들을 포함할 수 있으나, 일부 경우들에 있어서는 단지 중쇄들 만을 포함할 수 있는 카멜리드(camelids)들에서 자연적으로 발생하는 항체들과 같이 보다 적은 쇄들을 포함할 수 있다. 항체들은 전적으로 단일원천으로부터 유도되거나 또는 "키메라"가 되거나, 즉 이하에서 상세하게 설명되는 바와 같이 상기 항체의 서로 다른 부분들이 서로 다른 두 항체들로부터 유도될 수 있다. 상기 항원 결합 단백질들, 항체들 또는 결합 단편들은 하이브리도마들 내에서 재조합 DNA 기술들에 의하거나 또는 손상되지 않은 항체들의 효소개열 또는 화학개열에 의하여 생산될 수 있다. 달리 표시되지 않는 한, 용어 "항체"에는 2개의 전장의 중쇄들 및 2개의 전장의 경쇄들을 포함하는 항체들에 더하여 유도체들, 변종들, 단편들 및 그들의 돌연변이들이 포함되며, 그 예들이 이하에서 기술된다.
용어 "경쇄"에는 결합 특이성을 부여하기에 충분한 가변영역 시퀀스를 갖는 전장의 경쇄 및 그들의 단편들이 포함된다. 전장의 경쇄에는 가변영역 도메인 VL 및 불변영역 도메인(constant region domain) CL들이 포함된다. 상기 경쇄의 가변영역 도메인은 상기 폴리펩티드의 아미노-말단에 존재한다. 경쇄들에는 카파쇄(kappa chains) 및 람다쇄(lambda chains)들이 포함된다.
용어 "중쇄"에는 결합 특이성을 부여하기에 충분한 가변영역 시퀀스를 갖는 전장의 중쇄 및 그들의 단편들이 포함된다. 전장의 중쇄에는 가변영역 도메인 VH 및 3개의 불변영역 도메인 CH1, CH2 및 CH3들이 포함된다. 상기 VH 도메인은 상기 폴리펩티드의 아미노-말단에 존재하고, 그리고 상기 CH 도메인들은 상기 폴리펩티드의 카르복시-말단에 가장 근접하는 CH3와 함께 카르복실-말단에 존재한다. 중쇄들은 IgG(IgGl, IgG2, IgG3 및 IgG4 서브타입들을 포함), IgA(IgAl 및 IgA2 서브타입들을 포함), IgM 및 IgE들을 포함하여 임의의 아이소타입이 될 수 있다.
본 명세서에서 사용되는 바와 같은 항체 또는 면역글로블린쇄(중쇄 또는 경쇄)의 용어 "면역학적으로 기능적인 단편(immunologically functional fragment)"(또는 단순히 "단편")은 전장의 쇄 내에 존재하는 적어도 일부의 아미노산들을 결여하기는 하나 항원에 특이적으로 결합할 수 있는 항체의 부분(이 부분이 어떻게 수득되거나 또는 합성되는 지에 무관하게)을 포함하는 항원 결합 단백질이다. 이러한 단편들은 생물학적으로 활성이며, 이들은 목표 항원에 특이적으로 결합하거나 또는 손상되지 않은 항체들을 포함하여 주어진 에피토프에의 특이적인 결합을 위한 다른 항원 결합 단백질들과 경쟁할 수 있다. 하나의 관점에 있어서, 이러한 단편은 상기 전장의 경쇄 또는 중쇄 내에 존재하는 적어도 하나의 CDR을 보유하고, 그리고 일부 구체예들에 있어서는 단일의 중쇄 및/또는 경쇄 또는 그들의 부분을 포함할 수 있다. 이들 생물학적으로 활성인 단편들은 재조합 DNA 기술들에 의하여 생산되거나 또는 손상되지 않은 항체들을 포함하는 항원 결합 단백질들의 효소개열 또는 화학개열에 의하여 생산될 수 있다. 면역학적으로 기능적인 면역글로블린 단편들에는 Fab, Fab', F(ab')2, Fv, 도메인 항체들 및 단일쇄 항체들이 포함되나, 이들에 제한되는 것은 아니며, 인간, 생쥐(mouse), 집쥐(rat), 카멜리드 또는 토끼들이 포함되나 이들에 제한되지는 않는 임의의 포유동물 원천으로부터 유도될 수 있다. 본 명세서에서 기술된 상기 항원 결합 단백질들의 기능부분, 예를 들어 하나 또는 그 이상의 CDRs은 제2단백질 또는 소형분자들에 공유적으로 결합되어 이관능(bifunctional)의 치료적인 특성들 또는 연장된 혈청 반감기를 갖는 신체 내에서의 특정의 목표에 지향되는 치료제(therapeutic agent)를 생산할 수 있다.
"Fab 단편"은 하나의 경쇄 및 하나의 중쇄의 CH1과 가변영역들이 포함된다. Fab 분자의 상기 중쇄는 다른 중쇄 분자와 이황화결합(disulfide bond)을 형성하지 않을 수 있다.
"Fc" 영역은 항체의 상기 CH1 및 CH2 도메인들을 포함하는 2개의 중쇄 단편들을 포함한다. 상기 2개의 중쇄 단편들은 2개 또는 그 이상의 이황화결합들에 의하여 그리고 상기 CH3 도메인들의 소수성의 작용들에 의하여 서로 고정된다.
"Fab' 단편"은 하나의 경쇄 및 상기 VH 도메인과 상기 CH1 도메인 및 또한 상기 CH1 및 CH2 도메인들 사이의 영역들을 포함하는 하나의 중쇄의 일부를 포함하여 쇄간(interchain) 이황화결합이 2개의 Fab' 단편들의 2개의 중쇄들 사이에 형성되어 F(ab')2 분자를 형성하도록 한다.
"F(ab')2 단편"은 2개의 경쇄들 및 상기 CH1과 CH2 도메인들 사이의 상기 불변영역의 일부를 포함하는 2개의 중쇄들을 포함하여 상기 2개의 중쇄들 사이에 쇄간 이황화결합이 형성되도록 한다. 따라서 F(ab')2 단편은 상기 2개의 중쇄들 사이의 이황화결합에 의하여 서로 고정되는 2개의 Fab' 단편들로 이루어진다.
상기 "Fv 영역"은 상기 중쇄 및 경쇄들 둘 다로부터의 상기 가변영역들을 포함하기는 하나 상기 불변영역들은 결여하고 있다.
"단일쇄 항체들"은 상기 중쇄 및 경쇄 가변영역들이 유연한 링커(flexible linker)로 연결되어 단일의 폴리펩티드쇄를 형성하는 Fv 분자들이며, 이는 항원-결합 영역을 형성한다. 단일쇄 항체들은 국제공개공보 공개번호 제WO 88/01649호 및 미합중국 특허 제4,946,778호 및 동 제5,260,203호들에 상세하게 논의되어 있으며, 이들의 상세한 설명들을 참조로 인용한다.
"도메인 항체"는 단지 중쇄의 상기 가변영역과 경쇄의 상기 가변영역 만을 포함하는 면역학적으로 기능적인 면역글로블린 단편이다. 일부 경우들에 있어서, 둘 또는 그 이상의 VH 영역들은 펩티드 링커(peptide linger)와 공유적으로 결합되어 2가의 도메인 항체(bivalent domain antibody)를 형성한다. 2가의 도메인 항체의 상기 두 VH 영역들은 동일하거나 또는 서로 다른 항원들을 목표할 수 있다.
"2가의 항원 결합 단백질" 또는 "2가의 항체"는 2개의 항원결합사이트(antigen binding sites)들을 포함한다. 일부 경우들에 있어서, 상기 두 결합사이트들은 동일한 항원 특이성들을 갖는다. 2가의 항원 결합 단백질들 및 2가의 항체들은 양특이성이 될 수 있다. 이하를 참조하시오.
"다중특이성 항원 결합 단백질(multispecific antigen binding protein)" 또는 "다중특이성 항체"는 하나 이상의 항원 또는 에피토프를 목표하는 것이다.
"양특이성", "이중-특이성(dual-specific)" 또는 "이관능(bifunctional)" 항원 결합 단백질 또는 항체는 각각 2개의 서로 다른 항원결합사이트들을 갖는 잡종 항원 결합 단백질 또는 항체이다. 양특이성 항원 결합 단백질들 및 항체들은 다중특이성 항원 결합 단백질 또는 다중특이성 항체의 종들이며, 하이브리도마들의 융합 또는 Fab' 단편들의 연결을 포함하나 이들에 제한되지 않는 다양한 방법들에 의해 생산될 수 있다. 예를 들면, 송시빌라이와 라흐만(Songsivilai and Lachmann)의 1990, Clin. Exp. Immunol. 79:315-321; 코스텔니와 그의 동료들(Kostelny et al)의 1992, J. Immunol. 148:1547-1553들을 참조하시오. 양특이성 항원 결합 단백질 또는 항체의 상기 두 결합사이트들은 2개의 서로 다른 에피토프들에 결합될 수 있으며, 이는 동일하거나 또는 서로 다른 단백질 목표들 상에 존재할 수 있다.
용어 "중화 항원 결합 단백질(neutralizing antigen binding protein)" 또는 "중화 항체(neutralizing antibody)"는 각각 리간드에 결합하고, 상기 리간드의 그의 결합 짝(binding partner)에의 결합을 방해하고 그리고 달리는 상기 리간드의 그의 결합 짝에로의 결합을 야기하는 생물학적 반응을 간섭하는 항원 결합 단백질 또는 항체이다. 항원 결합 단백질, 예를 들면 항체 또는 면역학적으로 기능적인 그들의 단편의 결합 및 특이성을 평가함에 있어서, 과량의 항체가 상기 리간드에 결합되는 결합 짝의 양을 적어도 약 20%, 30%, 40%, 50%, 60%, 70%, 80%, 85%, 90%, 95%, 97%, 99% 또는 그 이상(시험관 내 경쟁적 결합 분석에서 측정된 바에 따라)로 감소시키는 경우에, 항체 또는 단편은 대체로 리간드의 그의 결합 짝에로의 결합을 억제할 수 있다. c-fms 항원 결합 단백질들의 경우에 있어서, 이러한 중화 분자는 c-fms가 CSF-1에 결합하는 능력을 감소시킬 수 있다. 일부 구체예들에 있어서, 상기 중화 항원 결합 단백질은 c-fms가 IL-34에 결합하는 능력을 억제한다. 다른 구체예들에 있어서, 상기 중화 항원 결합 단백질은 c-fms가 CSF-1 및 IL-34에 결합하는 능력을 억제한다.
동일한 에피토프에 대하여 경쟁하는 항원 결합 단백질들(예를 들면, 중화 항원 결합 단백질들 또는 중화 항체들)이 사용되는 경우에서 용어 "경쟁"은 시험 하의 상기 항원 결합 단백질(예를 들면, 항체 또는 면역학적으로 기능적인 그들의 단편)이 공통의 항원(예를 들면, c-fms 또는 그의 단편)에의 대조 항원 결합 단백질(예를 들면, 리간드 또는 대조 항체)의 특이적인 결합을 방해하거나 또는 저해하는 분석에 의해 결정되는 항원 결합 단백질들 사이의 경쟁을 의미한다. 예를 들면, 고상 직접 또는 간접 방사성면역분석법(solid phase direct or indirect radioimmunoassay ; RIA), 고상 직접 또는 간접 효소 면역분석법(solid phase direct or indirect enzyme immunoassay ; EIA), 샌드위치 경쟁 분석법(sandwich competition assay(예를 들면, 스탈리와 그의 동료들의 1983, Methods in Enzymology 9:242-253를 참조하시오); 고상 직접 비오틴-아비딘 EIA(커크랜드와 그의 동료들의 1986, J. Immunol. 137:3614-3619을 참조하시오), 고상 직접 표지분석법(solid phase direct labeled assay), 고상 직접 표지 샌드위치 분석법(solid phase direct labeled sandwich assay)(예를 들면, 하로우와 레인(Harlow and Lane)의 1988, Antibodies, A Laboratory Manual, Cold Spring Harbor Press을 참조하시오); I-125를사용하는 고상 직접 표지 RIA법(예를 들면, 모렐과 그의 동료들(Morel et al)의 1988, Molec. Immunol. 25:7-15를 참조하시오); 고상 직접 비오틴-아비딘 EIA법(예를 들면, 체웅과 그의 동료들(Cheung, et al.)의 1990, Virology 176:546-552); 및 직접 표지 RIA법(몰덴하우어와 그의 동료들(Moldenhauer et al.)의 1990, Scand. J. Immunol. 32:77-82) 등의 여러 형태들의 경쟁적 결합 분석(competitive binding assays)들이 사용될 수 있다. 전형적으로, 이러한 분석법들은 고체 표면에 결합하는 정제된 항원 또는 이들 중의 어느 하나를 포함하는 세포들, 표지되지 않은 시험 항원 결합 단백질 및 표지된 대조 항원 결합 단백질의 사용을 포함한다. 경쟁적 저해는 시험 항원 결합 단백질의 존재 중에서의 상기 고체 표면에 결합된 표지 또는 세포들의 양을 결정하는 것에 의하여 측정된다. 대개 상기 시험 항원 결합 단백질은 과량으로 존재한다. 경쟁적 분석에 의해 확인된 항원 결합 단백질들(경쟁하는 항원 결합 단백질들)에는 상기 대조 항원 결합 단백질들로서의 동일한 에피토프에 결합하는 항원 결합 단백질들 및 입체장애(steric hindrance)가 일어나도록 상기 대조 항원 결합 단백질에 의하여 결합된 상기 에피토프에 충분히 근접하는 인접하는 에피토프에 결합하는 항원 결합 단백질들이 포함된다. 경쟁적 결합을 결정하기 위한 방법들에 관한 별도의 상세한 설명들은 본 명세서의 실시예들에서 제공된다. 대개, 경쟁하는 항원 결합 단백질이 과량으로 존재하는 경우, 대조 항원 결합 단백질의 공통의 항원에의 특이적인 결합을 적어도 40%, 45%, 50%, 55%, 60%, 65%, 70% 또는 75%로 저해할 수 있다. 일부 경우들에 있어서, 결합은 적어도 80%, 85%, 90%, 95% 또는 97% 또는 그 이상으로 저해된다.
용어 "항원"은 항원 결합 단백질(예를 들면, 항체 또는 면역학적으로 기능적인 그들의 단편을 포함) 등과 같은 선택적 결합제(selective binding agent)에 의해 결합될 수 있고, 그리고 부가적으로 동물들 내에서 사용되어 상기 항원에의 결합이 가능한 항체들을 생산할 수 있는 분자 또는 분자의 부분을 의미한다. 항원은 서로 다른 항원 결합 단백질들, 예를 들면, 항체들과 상호작용할 수 있는 하나 또는 그 이상의 에피토프들을 포함할 수 있다.
용어 "에피토프"는 항원 결합 단백질(예를 들면, 항체)에 의해 결합되는 분자의 부분이다. 상기 용어는 항체 등과 같은 항원 결합 단백질에 또는 T-세포 수용체(T-cell receptor)에 특이적으로 결합할 수 있는 임의의 결정요인(determinant)을 포함한다. 에피토프는 인접적이거나 또는 비-인접적(예를 들면, 폴리펩티드 내에서, 상기 폴리펩티드 시퀀스 내의 다른 하나에 인접하지 않으나 상기 분자의 정황 내에서 상기 항원 결합 단백질에 의하여 결합되는 아미노산 잔기들)일 수 있다. 특정의 구체예들에 있어서, 에피토프들은 모방체가 될 수 있으며, 여기에서 이들은 상기 항원 결합 단백질을 생성하는 데 사용된 에피토프와 유사한 3차원의 구조를 포함하나, 그러나 상기 항원 결합 단백질을 생성하는 데 사용된 에피토프 내에서 발견된 아미노산 잔기들의 전부 포함하지 않거나 또는 단지 일부만을 포함한다. 종종, 에피토프들은 단백질들 상에 존재하나, 일부 경우들에 있어서는 핵산들 등과 같은 다른 종류들의 분자들 상에 존재할 수도 있다. 에피토프 결정요인들에는 아미노산, 당측쇄들, 포스포릴기 또는 설포닐기들 등과 같은 화학적으로 활성인 표면 그룹들(chemically active surface groupings)이 포함될 수 있으며, 특정의 3차원의 구조상 특징들 및/또는 특정의 하전특성들을 가질 수 있다. 대체로, 특정의 목표 항원에 특이적인 항체들은 단백질들 및/또는 거대분자(macromolecules)들의 복잡한 혼합물 내의 상기 목표 항원 상의 에피토프를 우선적으로 인식할 수 있을 것이다.
용어 "동일성"은 시퀀스들의 정렬(aligning) 및 비교(comparing)에 의해 결정되는 바와 같이 둘 또는 그 이상의 폴리펩티드 분자들 또는 둘 또는 그 이상의 핵산 분자들의 시퀀스들 사이의 관계를 의미한다. "동일성 백분율(percent identity)"은 비교되는 분자들 내의 아미노산들 또는 뉴클레오티드들 사이의 동일한 잔기들의 백분율을 의미하며, 비교되는 상기 분자들의 가장 작은 크기에 기초하여 계산된다. 이들 계산들을 위하여는, 정렬들에서의 갭(gaps)들(있는 경우)은 특정의 수학적 모델 또는 컴퓨터 프로그램(즉, "알고리즘(algorithm)")에 의해 접근되어야만 한다. 상기 정렬된 핵산들 또는 폴리펩티드들의 동일성을 계산하는 데 사용될 수 있는 방법들에는 컴퓨터 상 분자생물학(Computational Molecular Biology, (Lesk, A. M., ed.), 1988, New York: Oxford University Press); 바이오컴퓨팅 인포매틱스 및 게놈 프로젝트(Biocomputing Informatics and Genome Projects, (Smith, D. W., ed.), 1993, New York: Academic Press); 시퀀스 데이터의 컴퓨터 분석 1부(Computer Analysis of Sequence Data, Part I, (Griffin, A. M., and Griffin, H. G., eds.), 1994, New Jersey: Humana Press); 본 힌제 지(von Heinje, G.)의 1987, Sequence Analysis in Molecular Biology, New York: Academic Press; 시퀀스 분석 프라이머(Sequence Analysis Primer, (Gribskov, M. and Devereux, J., eds.), 1991, New York: M. Stockton Press); 및 카릴로와 그의 동료들(Carillo et al)의 1988, SIAMJ. Applied Math. 48:1073들에 기술된 방법들이 포함된다.
동일성 백분율의 계산에 있어서, 비교되는 상기 시퀀스들은 상기 시퀀스들 사이에서 가장 큰 매치(match)를 부여하는 방법으로 정렬된다. 동일성 백분율을 결정하는 데 사용되는 상기 컴퓨터 프로그램은 GCG 프로그램 패키지이며, 이는 GAP을 포함한다(데베레욱스와 그의 동료들(Devereux et al)의 1984, Nucl. Acid Res. 12:387; 제네틱 컴퓨터 그룹(Genetics Computer Group, University of Wisconsin, Madison, WI). 상기 컴퓨터 알고리즘 GAP는 시퀀스 동일성 백분율이 결정되어야 할 두 개의 폴리펩티드들 또는 폴리뉴클레오티드들을 정렬하는 데 사용된다. 상기 시퀀스들은 그들 개개 아미노산 또는 뉴클레오티드의 최적의 매칭을 위하여 정렬된다(상기 알고리즘에 의해 결정된 바와 같은 "매칭된 스판(matched span"). 갭 오프닝 페널티(gap opening penalty ; 평균 항의 3X로 통상적으로 계산됨, 여기에서 "평균 항(average diagonal)"은 사용되고 있는 비교 매트릭스의 대각선의 평균이고; "항"은 특정 비교 매트릭스에 의해 각 완전 아미노산 매치에 할당된 스코어 또는 수이다) 및 갭 연장 페널티(gap extension penalty ; 통상적으로 갭 오프닝 페널티의 1/10배임) 뿐만 아니라 PAM 250 또는 BLOSUM 62 등과 같은 비교 매트릭스가 상기 알고리즘과 함께 이용된다. 특정의 구체예에 있어서, 표준 비교 매트릭스 (PAM250 비교 매트릭스에 대하여는 데이호프와 그의 동료들(Dayhoff et al.)의 1978, Atlas of Protein Sequence and Structure 5:345-352를; BLOSUM 62 비교 매트릭스에 대하여는 헤니코프와 그의 동료들(Henikoff et al)의 1992, Proc. Natl. Acad. Sci. U.S.A. 89:10915-10919를 참조하시오)가 또한 상기 알고리즘에 의하여 사용된다.
상기 GAP 프로그램을 사용하는 폴리펩티드들 또는 뉴클레오티드 시퀀스들에 대한 동일성 백분율을 결정하기 위한 추천되는 파라미터들은 다음과 같다:
알고리즘: 니들만과 그의 동료들(Needleman et al)의 1970, J. MoI. Biol. 48:443-453;
비교 매트릭스: 헤니코프와 그의 동료들(Henikoff et al.)의 위의 1992 문헌으로부터의 BLOSUM 62;
갭 페널티: 12(그러나 엔드 갭들에 대하여는 페널티 없음)
갭 길이 페널티: 4
유사성의 문턱값(Threshold of Similarity): 0
2개의 아미노산 시퀀스들의 정렬을 위한 특정의 정렬 계획들은 상기 두 시퀀스들의 단지 짧은 영역의 매칭이라는 결과를 가져올 수 있으며, 비록 상기 두 전장의 시퀀스들 사이에서 뚜렷한 관계가 없는 경우에도 이러한 작은 정렬된 영역은 매우 높은 시퀀스 동일성을 가질 수 있다. 따라서, 선택된 정렬 방법(GAP 프로그램)은 원하는 경우 상기 목표 폴리펩티드의 적어도 50개의 인접하는 아미노산들에 걸치는 정렬이라는 결과를 낳도록 조정될 수 있다.
본 명세서에서 사용된 바와 같이, "대체로 순수한(substantially pure)"은 기술된 분자의 종들이 우월하게 존재하는 종들인 즉, 몰 기반에서(molar basis) 동일한 혼합물 내에서 임의의 다른 개개 종들 보다 훨씬 더 풍부하다는 것이다. 특정의 구체예들에 있어서, 대체로 순수한 분자는 대상 종들이 존재하는 전체 거대분자상의 종들 중의 적어도 50%(몰 기반으로)를 포함하는 조성물이다. 다른 구체예들에 있어서, 대체로 순수한 조성물은 상기 조성물 내에 존재하는 모든 거대분자상의 종들의 적어도 80%, 85%, 90%, 95% 또는 99%를 포함할 수 있다. 다른 구체예들에 있어서, 상기 대상 종들은 필수적인 동종성으로 정제되며, 여기에서 오염시키는 종들은 상기 조성물 내에서 통상의 검출 방법들로는 검출되지 않으며, 따라서 상기 조성물이 단일의 검출가능한 거대분자상 종들로 이루어진다.
용어 "치료(treating)"는 완화(abatement); 차도(remission); 환자가 증후군들 또는 손상, 병적 측면 또는 상태들의 경감(diminishing)이나 또는 보다 더 견딜 수 있도록 만드는 것; 악화 또는 감소의 속도의 저하; 악화의 종말점을 덜 악화되게 만드는 것; 환자의 신체적 또는 정신적 웰빙(well-being)을 개선하는 것들을 포함하여 임의의 목적 또는 대상체 인자들을 포함하여 손상, 병적 측면 또는 상태의 치료 또는 완화에서 임의의 성공의 징후를 의미한다. 증후군들의 치료 또는 완화는 신체검사, 신경정신병학적 검사(neuropsychiatric exams) 및/또는 정신병학적 평가들의 결과들을 포함하여 목적하는 또는 대상의 파라미터들에 기초할 수 있다. 예를 들면, 본 명세서에 존재하는 특정의 방법들이 암의 발생의 감소, 암의 차도 및/또는 암 또는 염증성 질병과 연관된 증후군들의 완화에 의하여 성공적으로 암을 치료한다.
"유효량"은 대체로 증후군들의 심각성 및/또는 빈도들을 감소시키거나, 상기 증후군들 및/또는 내재하는 원인들을 제거하거나, 증후군들 및/또는 그들의 내재하는 원인의 발생을 예방하거나 및/또는 암으로부터 또는 암과 연관되는 손상을 개선 또는 복원하기에 충분한 양이다. 일부 구체예들에 있어서, 상기 유효량은 치료적 유효량 또는 예방적 유효량이다. "치료적 유효량"은 질병 상태(예를 들면, 암) 또는 증후군들, 특히 질병 상태와 연관된 상태 또는 증후군들을 치료하거나 달리 질병 상태 또는 상기 질병과 관련된 임의의 다른 원치않는 증후군의 진행을 예방, 저해, 지연 또는 가역 등등을 시키기에 충분한 양이다. "예방적 유효량"은 대상체에 투여되는 경우, 목적하는 예방적인 효과 예를 들면 암의 개시(onset) 또는 재발의 예방 또는 지연 또는 암 또는 암 증후군들의 개시(또는 재발)의 가능성의 감소시킬 수 있는 약제학적 조성물의 양이다. 완전한 치료 또는 예방적 효과는 1회의 투여량(dose)의 투여에 의해 반드시 일어난다고 할 수는 없으며, 단지 일련의 투여량들의 투여 이후에 일어날 수 있다. 따라서, 치료적 또는 예방적 유효량은 1회 또는 그 이상의 투여에서 투여될 수 다.
"아미노산"은 당해 기술분야에서 그의 통상의 의미를 포함한다. 20개의 자연적으로 발생하는 아미노산들 및 그들의 약어들은 통상의 용례를 따른다. 본 명세서에서 임의의 목적을 위한 참조로 인용되는 Immunology-A Synthesis, 2nd Edition, (E. S. Golub and D. R. Green, eds.), Sinauer Associates: Sunderland, Mass. (1991)을 참조하시오. 20개의 통상의 아미노산들의 입체이성질체들(예를 들면, D-아미노산), [알파]-,[알파]-이치환 아미노산들 등과 같은 비자연의 아미노산들, N-알킬아미노산들 및 다른 비통상적인 아미노산들이 폴리펩티드들의 구성요소들로서 또한 적절할 수 있으며, 상기 문구 "아미노산" 내에 포함된다. 비통상적인 아미노산들의 예들에는 4-히드록시프롤린, [감마]-카르복시글루타메이트, [입실론]-N,N,N-트리메틸리신, [입실론]-N-아세틸리신, O-포스포세린, N-아세틸세린, N-포밀메티오닌, 3-메틸히스티딘, 5-히드록시리신, [시그마]-N-메틸아르기닌 및 다른 유사한 아미노산들 및 이미노산(imino acids)(예를 들면, 4-히드록시프롤린)들이 포함된다. 본 명세서에서 사용되는 상기 폴리펩티드 표기에 있어서, 표준 용례 및 관례에 따라 상기 좌수방향이 아미노 말단 방향이고, 그리고 상기 우수방향이 상기 카르복시 말단 방향이다.
개요
인간 c-fms(hc-fms)를 포함하여 c-fms 단백질을 결합하는 항원-결합 단백질들이 제공된다. 제공되는 상기 항원 결합 단백질들은 본 명세서에서 기술된 바와 같이 그 안으로 하나 또는 그 이상의 상보적 결합 영역들(CDRs)이 매립되거나 및/또는 결합된 폴리펩티드들이다. 일부 항원 결합 단백질들에 있어서, 상기 CDR들은 "골격" 영역 내로 매립되며, 이는 상기 CDR(들)의 상기 적절한 항원 결합 특헝들이 달성되도록 상기 CDR(들)을 지향시킨다. 대체로, 제공되는 항원 결합 단백질들은 CSF-1과 c-fms 사이의 상호작용을 간섭하거나 차단하거나 감소시키거나 또는 조정할 수 있다.
본 명세서에서 기술된 특정의 항원 결합 단백질들은 항체들이거나 또는 항체들로부터 유도된다. 특정의 구체예들에 있어서, 상기 항원 결합 단백질들의 상기 폴리펩티드 구조는 모노클로날 항체들, 양특이성 항체들, 미니바디(minibodies)들, 도메인 항체들, 합성 항체들(때때로 여기에서는 "항체 모방체들"이라고도 언급됨), 키메라 항체들, 인간화된 항체들, 인간 항체들, 항체 융합물(때때로 여기에서는 "항체 콘쥬게이트(antibody conjugates)"라고도 언급됨) 및 이들의 단편들을 포함하나 이들에 제한되지는 않는 항체들에 기초하고 있다. 다양한 구조들이 이하에서 더욱 기술된다.
본 명세서에서 제공된 상기 항원 결합 단백질들은 c-fms, 특히 인간 c-fms의 세포외 도메인(extracellular domain)에 결합한다는 것이 입증되었다. 이하의 구체예들에서 더 기술되는 바와 같이, 특정의 항원 결합 단백질들은 다수의 다른 항-c-fms 항체들에 의해 결합되어 있는 그들로부터의 다른 에피토프들에 결합한다는 것이 시험으로 밝혀졌다. 제공되는 상기 항원 결합 단백질들은 CSF-1과 경쟁하고 그리고 그에 의하여 CSF-1이그의 수용체에 결합하는 것을 방해한다. 특정의 구체예들에 있어서, 항원 결합 단백질들은 IL-34 및 c-fms들 사이의 결합을 저해한다. 다른 구체예들에 있어서, 상기 항원 결합 단백질들은 c-fms의 CSF-1 및 IL-34 둘 다에 결합하는 능력을 저해한다. 따라서, 본 명세서에서 제공되는 상기 항원 결합 단백질들은 c-fms 활성을 저해하는 능력이 있다. 특히, 이들 에피토프들에 결합하는 항원 결합 단백질들은 하나 또는 그 이상의 하기의 활성들, 즉, 저해, 그 중에서도 c-fms 자가인산화, c-fms 신호 변환 경로들의 유도, c-fms 유도 세포 성장, 종양 또는 종양의 스트로마 내에서의 종양 연관 마크로파지들의 단핵구 주화성 축적, CSF-1 결합에 의한 c-fms에 의해 유도되는 종양-증식 인자들의 생산 및 다른 생리학적 영향들의 저해들을 가질 수 있다. 본 명세서에서 기술되는 상기 항원 결합 단백질들은 다양한 유용성을 갖는다. 예를 들면, 상기 항원 결합 단백질들 중의 일부들은 특이적 결합 분석법, c-fms, 특히 hc-fms 또는 그 의 리간드들의 친화도 정제 및 c-fms 활성의 다른 길항근(antagonists)들을 확인하는 스크리닝분석법들에서 유용하다. 상기 항원-결합 단백질들의 일부는 CSF-1의 c-fms에의 결합을 저해하거나 또는 c-fms의 자가인산화를 저해하는 데 유용하다.
상기 항원-결합 단백질들은 본 명세서에서 설명되는 바와 같이 다양한 치료적용례들에서 사용될 수 있다. 예를 들면, 특정의 c-fms 항원-결합 단백질들은 이하에서 더 기술되는 바와 같이 환자에서의 단핵구 주화성의 감소, 종양들 내로의 단핵구 이동의 저해, 종양 내에서의 종양 관련 마크로파지의 축적의 저해 또는 신생혈관형성(angiogenesis)의 저해 등과 같은 c-fms와 연관된 상태들을 치료하는 데 유용하다. 특정의 구체예들에 있어서, 상기 항원 결합 단백질들은 TAMs의 종양 성장, 진행 및/또는 전이를 촉진하는 능력을 저해한다. 게다가, 상기 종양 세포들이 자체적으로 발현되고 그리고 c-fms를 사용하는 경우들에 있어서, c-fms에 uf합하는 항체는 그들의 성장/생존을 저해할 수 있다. 상기 항원 결합 단백질들에 대한 다른 용도들에는 예를 들면 c-fms-연관 질병들 또는 상태들의 진단 및 c-fms의 존재 또는 부재를 결정하기 위한 스크리닝 분석법들이 포함된다. 본 명세서에서 기술되는 상기 항원 결합 단백질들의 일부는 c-fms 활성과 연관되는 결과들, 증후군들 및/또는 병적 측면을 치료하는 데 유용하다. 이들에는 암 악액질(cancer cachexia)과 마찬가지로 여러 형태들의 암 및 염증성 질병들이 포함되나 이들에 제한되는 것은 아니다. 일부 구체예들에 있어서, 상기 항원 결합 단백질들은 여러 골장애(bone disorders)들을 치료하는 데 사용될 수 있다.
C-fms
콜로니-자극인자 1(CSF-1)은 단핵의 식세포 계열(mononuclear phagocyte lineages)들의 생존, 증식 및 분화를 촉진한다. CSF-1은 상기 세포-표면 c-fms 수용체에의 결합에 의하여 그의 활성들을 발휘하여 수용체 c-fms 키나아제 및 후속하는 세포간 신호들의 폭포에 의한 자가인산화라는 결과를 가져온다.
용어 "c-fms", "c-fms 수용체", "인간 c-fms" 및 "인간 c-fms 수용체"들은 CSF-1을 포함하나 이에 제한되지 않는 리간드에 결합하고 그 결과 상기 세포 내에서의 신호 전달 경로를 개시시키는 세포 표면 수용체를 의미한다. 일부 구체예들에 있어서, 상기 수용체는 IL-34 또는 CSF-1과 IL-34 둘 다에 결합할 수 있다. 본 명세서에서 기술된 상기 항원 결합 단백질들은 c-fms, 특히 인간 c-fms에 결합한다. 인간 c-fms 아미노산 시퀀스의 예시적인 세포외 도메인은 SEQ ID NO: 1에서 묘사된 것이다. 이하에서 기술되는 바와 같이, c-fms 단백질들은 또한 단편들을 포함할 수 있다.본 명세서에서 사용되는 바와 같이, 상기 용어들은 수용체, 특히 CSF-1에 특이적으로 결합하는 인간 수용체를 의미하는 것으로 상호교환적으로 사용된다.
본 명세서에서 사용되는 바와 같은 용어 인간 c-fms(h-c-fms) 수용체는 또한 돌연변이 A245S, V279M 및 H362R들을 포함하여 자연적으로 발생하는 대립형질(alleles)들을 포함한다. 상기 용어 c-fms는 또한 상기 c-fms 아미노산 시퀀스의 전사후 변형(post-translational modifications)들을 포함한다. 예를 들면, 인간 c-fms(상기 수용체의 20-512 잔기들)의 상기 세포외 도메인(ECD)은 상기 시퀀스 내에 11개의 가능한 N-연결 글리코실화 사이트들을 갖는다. 따라서, 상기 항원 결합 단백질들은 상기 위치들 중의 하나 또는 그 이상에서 글리코실화된 단백질에 결합되거나 또는 그로부터 생성될 수 있다.
상기 c-fms 신호 전달 경로는 조직 마크로파지 분포(tissue marcrophage populations)들의 만성 활성(chronic activation)을 포함하는 인간 병적 측면들의 수를 증가(up-regulated)시킨다. CSF-1 생산에서의 증가는 또한 염증성 장 질환 등과 같은 여러 염증성 질병들에서 보여지는 조직 마크로파지들의 축적과 연관이 있다. 게다가, 여러 종양 형태들의 성장은 암세포들 및/또는 종양 스트로마 내에서의 CSF-1 및 c-fms 수용체의 과발현과 연관된다.
C-fms 수용체 항원 결합 단백질들
c-fms의 활성을 조절하는 데 유용한 다양한 선택적 결합제들이 제공된다. 이들 약품들에는, 예를 들면, 항원 결합 도메인(예를 들면, 단일쇄 항체들, 도메인 항체들, 면역접착제(inmmunoadhesions) 및 항원 결합 영역을 갖는 폴리펩티드들)을 포함하고 그리고 c-fms 폴리펩티드, 특히 인간 c-fms에 특이적으로 결합하는 항원 결합 단백질들이 포함된다. 상기 약품들의 일부는, 예를 들면, CSF-1의 c-fms에의 결합을 저해하는 데 유용하며, 따라서 c-fms 신호화와 연관된 하나 또는 그 이상의 활성들을 저해하거나, 간섭하거나 또는 조절하는 데 사용될 수 있다. 특정의 구체예들에 있어서, 상기 항원 결합 단백질들은 IL-34와 c-fms 간의 결합을 저해하는 데 사용될 수 있다. 일부 구체예들에 있어서, 상기 항원 결합 단백질들은 c-fms의 CSF-1 및 IL-34 둘 다에 결합하는 능력을 간섭한다.
대체로, 전형적으로 제공되는 상기 항원 결합 단백질들은 본 명세서(예를 들면, 1, 2, 3, 4, 5 또는 6)에서 기술되는 하나 또는 그 이상의 CDRs들을 포함한다. 일부 경우들에서, 상기 항원 결합 단백질은 (a) 폴리펩티드 구조 및 (b) 상기 폴리펩티드 구조 내로 삽입되거나 및/또는 결합되는 하나 또는 그 이상의 CDRs들을 포함한다. 상기 폴리펩티드 구조는 다양한 서로 다른 형태들을 취할 수 있다. 예를 들면, 자연적으로 발생하는 항체, 또는 그의 단편이나 변형들의 골격이 되거나 또는 그를 포함하는 것이 되거나 또는 자연에서 완전히 합성되는 것이 될 수 있다. 여러 폴리펩티드 구조들의 예들을이하에서 기술한다.
특정의 구체예들에 있어서, 상기 항원 결합 단백질들의 상기 폴리펩티드 구조는 항체이거나 또는 모노클로날 항체들, 양특이성 항체들, 미니바디들, 도메인 항체들, 합성 항체들(때때로 여기에서는 "항체 모방체들"이라고도 언급됨), 키메라 항체들, 인간화된 항체들, 항체 융합물(때때로 여기에서는 "항체 콘쥬게이트(antibody conjugates)"라고도 언급됨) 및 각 이들의 부분들이나 단편들로부터 유도되는 것이나 이들에 제한되지는 않는다. 일부 경우들에 있어서, 상기 항원 결합 단백질은 항체(예를 들면, Fab, Fab', F(ab')2 또는 scFv)의 면역학적 단편이다. 상기 다양한 구조들을 이하에서 기술하고 그리고 정의한다.
본 명세서에서 제공되는 바와 같은 상기 항원 결합 단백질들 중의 특정의 것은 인간 c-fms에 특이적으로 결합한다. 특정의 구체예에 있어서, 상기 항원 결합 단백질은 SEQ ID NO: 1의 아미노산 시퀀스를 갖는 인간 c-fms 단백질에 특이적으로 결합한다.
상기 항원 결합 단백질이 치료적인 응용예들에 대하여 사용되는 구체예들에 있어서, 항원 결합 단백질은 c-fms의 하나 또는 그 이상의 생물학적 활성들을 저해하거나 간섭하거나 또는 조절할 수 있다. 이 경우에 있어서, 과량의 항체가 CSF-1에 결합되거나 또는 그의 역인 인간 c-fms의 양을 적어도 약 20%, 30%, 40%, 50%, 60%, 70%, 80%, 85%, 90%, 95%, 97%, 99% 또는 그 이상(예를 들면, 시험관 내 경쟁적 결합 분석법에서의 결합을 측정하는 것에 의함)으로 감소시키는 경우에 인간 c-fms가 CSF-1에 특이적으로 결합하거나 및/또는 결합을 저해한다. c-fms는 많은 뚜렷한 생물학적 효과들을 가지며, 이는 서로 다른 세포형들 내에서 여러 서로 다른 분석법들로 측정될 수 있으며, 이러한 분석법들의 예들을 이하에서 제공한다.
자연적으로 발생하는 항체 구조
제공되는 상기 항원 결합 단백질들의 일부는 전형적으로 자연적으로 발생하는 항체들과 연관되는 구조를 갖는다. 비록 포유동물들의 일부 종들이 또한 단지 단일의 중쇄만을 갖는 항체들을 생산하기는 함에도 불구하고, 이들 항체들의 구조상 단위들은 전형적으로 하나 또는 그 이상의 테트라머(tetramers)들을 포함하며, 각각은 폴리펩티드쇄들의 2개의 동일한 쿠플레(couplets)들로 이루어진다. 전형적인 항체에 있어서, 쿠플레의 각 쌍은 하나의 전장의 "경"쇄(특정의 구체예들에 있어서는 대략 25kDa) 및 하나의 전장의 "중"쇄(특정의 구체예들에 있어서는 대략 50 내지 70kDa)를 포함한다. 각 개별 면역글로블린쇄는 여러 "면역글로블린 도메인들"로 이루어지며, 각각은 대략 90 내지 110개의 아미노산들로 이루어지며 특이적인 중첩 패턴(folding pattern)을 발현한다. 이들 도메인들은 그의 폴리펩티드들이 구성되는 기본단위(basic units)들이다. 각 쇄의 상기 아미노-말단 부분은 전형적으로 항원 인식을 책임지는 가변영역을 포함한다. 상기 카르복시-말단 부분은 상기 쇄의 다른 단부보다 더 진화적으로 보존되며, "불변영역" 또는 "C 영역"으로 칭하여진다. 인간 경쇄들은 대체로 카파경쇄 및 람다경쇄들로 분류되며, 이들 각각은 하나의 가변 도메인과 하나의 불변 도메인을 포함한다. 중쇄들은 전형적으로 뮤(mu), 델타(delta), 감마(gamma), 알파(alpha) 또는 입실론(epsilon)으로 분류되며, 이들은 항체의 아이소타입을 각각 IgM, IgD, IgG, IgA 및 IgE로 정의한다. IgG는 IgGl, IgG2, IgG3 및 IgG4를 포함하여 여러 서브타입들을 가지나, 이들에 제한되는 것은 아니다. IgM 서브타입들에는 IgM 및 IgM2들이 포함된다. IgA 서브타입들에는 IgA1 및 IgA2들이 포함된다. 인간에 있어서, 상기 IgA 및 IgD 아이소타입들은 4개의 중쇄들 및 4개의 경쇄들을 포함하고; 상기 IgG 및 IgE 아이소타입들은 2개의 중쇄들 및 2개의 경쇄들을 포함하고; 그리고 상기 IgM 아이소타입은 5개의 중쇄들 및 5개의 경쇄들을 포함한다. 상기 중쇄 C 영역은 전형적으로 효과기 기능(effector function)에 책임이 있는 하나 또는 그 이상의 도메인들을 포함한다. 중쇄 불변영역 도메인들의 수는 아이소타입에 의존될 수 있다. 예를 들면, IgG 중쇄들 각각은 CH1, CH2 및 CH3들로 알려진 3개의 C 영역 도메인들을 포함한다. 제공되는 상기 항체들은 이들 아이소타입들 및 서브타입들 중의 임의의 것을 가질 수 있다. 특정의 구체예들에 있어서, 상기 c-fms 항체는 IgGl, IgG2 또는 IgG4 서브타입들 중의 것이다.
전장의 경쇄 및 중쇄들에 있어서, 상기 가변 및 불변영역들은 또한 대략 10 또는 그 이상의 아미노산들의 "디(D)" 영역들을 포함하는 중쇄와 함께 대략 12 또는 그 이상의 아미노산들의 "제이(J)" 영역에 의해 결합된다. 예를 들면, 기초 면역학(Fundamental Immunology, 2nd ed., Ch. 7 (Paul, W., ed.) 1989, New York: Raven Press (전체 목적들을 위하여 그의 전체로서 본 명세서에 참고로 인용됨)을 참조하시오. 각 경쇄/중쇄 쌍의 상기 가변영역들은 전형적으로 상기 항원결합사이트를 형성한다.
예시적인 c-fms 모노클로날 항체의 IgG2 중쇄 불변 도메인의 하나의 구체예는 하기 아미노산 시퀀스를 갖는다:
ASTKGPSVFPLAPCSRSTSESTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSNFGTQTYTCNVDHKPSNTKVDKTVERKCCVECPPCPAPPVAGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVQFNWYVDGVEVHNAKTKPREEQFNSTFRVVSVLTVVHQDWLNGKEYKCKVSNKGLPAPIEKTISKTKGQPREPQVYTLPPSREEMTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPMLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK* (SEQ. ID NO: 2 ; 별표는 정지 코돈에 대응한다).
예시적인 c-fms 모노클로날 항체의 카파경쇄 불변 도메인의 하나의 구체예는 하기 아미노산 시퀀스를 갖는다:
RTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC* (SEQ ED NO: 3 ; 별표는 정지 코돈에 대응한다).
면역글로블린쇄들의 가변영역들은 종종 "보족결정영역들" 또는 CDRs라 불리우는 3개의 고도가변영역(hypervariable regions)들에 의해 결합되는 상대적으로 보존되는 골격영역(FR)들을 포함하여 대체로 동일한 전체 구조를 나타낸다. 앞서 언급된 각 중쇄/경쇄쌍의 2개의 쇄들로부터의 상기 CDRs들은 상기 골격영역에 의해 정렬되어 상기 목표 단백질(예를 들면, c-fms) 상의 특이적인 에피토프와 특이적으로 결합하는 구조를 형성한다. N-말단으로부터 C-말단까지, 자연적으로-발생하는 경쇄 및 중쇄 가변영역들 둘 다는 전형적으로 이들 요소들의 다음의 순서와 일치한다: FRl, CDRl, FR2, CDR2, FR3, CDR3 및 FR4. 번호부여 시스템(numbering system)은 이들 도메인들의 각각에서의 위치들을 점유하는 아미노산들에 번호를 부여하기 위하여 고안되었다. 이러한 번호부여 시스템은 면역학적 대상의 단백질들의 카바트 시퀀스(Kabat Sequences of Proteins of Immunological Interest (1987 and 1991, NIH, Bethesda, MD) 또는 초치아와 레스크(Chothia & Lesk)의 1987, J. MoI. Biol. 196:901-917; 초치아와 그의 동료들(Chothia et al)의 1989, Nature 342:878-883에서 정의된다.
본 명세서에서 제공되는 상기 여러 중쇄 및 경쇄 가변영역들을 표 2(예시적인 VH 및 VL쇄들)에 묘사하였다. 이들 가변영역들 각각은 상기 중쇄 및 경쇄 불변영역들에 부착되어 각각 완전한 항체 중쇄 및 경쇄를 형성한다. 또한, 그렇게 생성된 중쇄 및 경쇄 시퀀스들 각각은 결합되어 완전한 항체 구조를 형성한다. 본 명세서에서 제공된 상기 중쇄 및 경쇄 가변영역들은 또한 이하에서 기술되는 상기 예시적인 시퀀스들과는 다른 시퀀스들을 갖는 다른 불변 도메인들에 부착될 수 있다.
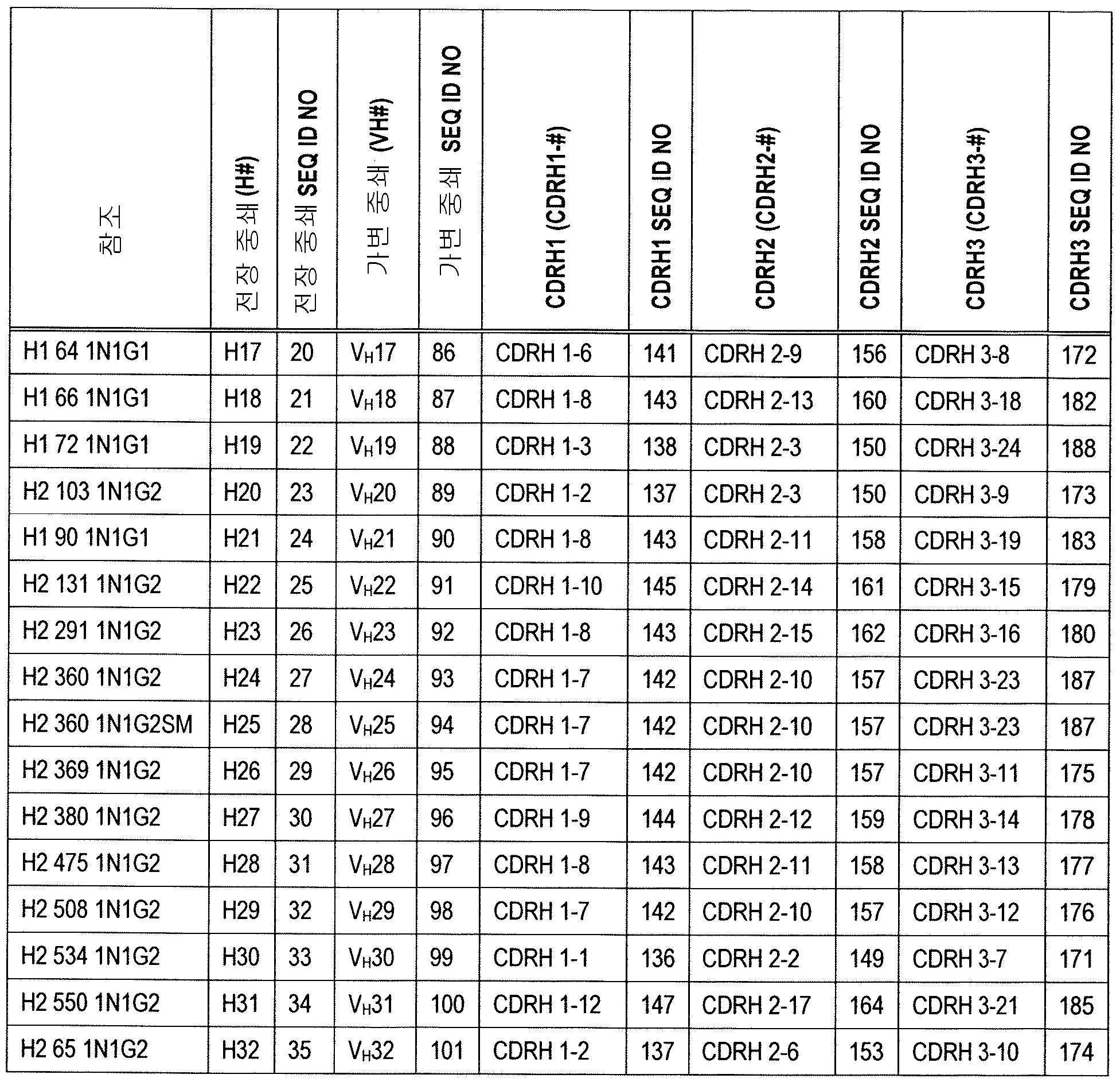
제공되는 상기 항체들의 상기 전장의 경쇄 및 중쇄들의 일부의 특정의 예들 및 그들의 대응하는 아미노산 시퀀스들을 표 1(예시적인 중쇄 및 경쇄들)에 요약하였다.
[표 1-1]
[표 1-2]
[표 1-3]
[표 1-4]
[표 1-5]
[표 1-6]
[표 1-7]
[표 1-8]
[표 1-9]
[표 1-10]
[표 1-11]
다시, 표 1에 개시된 상기 예시적인 중쇄(Hl, H2, H3 등)들은 표 1에 나타낸 상기 예시적인 경쇄들과 조합하여 항체를 형성할 수 있다. 이러한 조합들의 구체예들에는 L1 내지 L34들 중의 임의의 것과 결합하는 H1; L1 내지 L34들 중의 임의의 것과 결합하는 H2; L1 내지 L34들 중의 임의의 것과 결합하는 H3; 등등이 포함된다. 일부 경우들에 있어서, 상기 항체들은 표 1에 개시된 것들로부터의 적어도 하나의 중쇄 및 하나의 경쇄를 포함한다. 일부 경우들에 있어서, 상기 항체들은 표 1에 개시된 2개의 서로 다른 중쇄들 및 2개의 서로 다른 경쇄들을 포함한다. 다른 경우들에 있어서, 상기 항체들은 2개의 동일한 경쇄들 및 2개의 동일한 중쇄들을 포함한다. 예시로서, 항체 또는 면역학적으로 기능적인 단편에는 표 1에 개시된 바와 같은 2개의 H1 중쇄들 및 2개의 L1 경쇄들, 또는 2개의 H2 중쇄들 및 2개의 L2 경쇄들, 또는 2개의 H3 중쇄들 및 2개의 L3 경쇄들 및 경쇄들의 쌍들과 중쇄들의 쌍들의 다른 유사한 조합들이 포함될 수 있다.
제공되는 다른 항원 결합 단백질들은 표 1에 나타낸 중쇄 및 경쇄들의 조합에 의해 형성되는 항체들의 변종들이며, 각각 이들 쇄들의 상기 아미노산 시퀀스들에 대하여 적어도 70%, 75%, 80%, 85%, 90%, 95%, 97% 또는 99%의 동일성을 갖는 경쇄 및/또는 중쇄들을 포함한다. 일부 경우들에 있어서, 이러한 항체들은 적어도 하나의 중쇄 및 하나의 경쇄를 포함하는 반면에, 다른 경우들에서는, 상기 변종 형태들은 2개의 동일한 경쇄 및 2개의 동일한 중쇄들을 포함한다.
항체들의 가변 도메인들
하기의 표 2에서 나타낸 바와 같은 VH1, VH2, VH3, VH4, VH5, VH6, VH7, VH8, VH9, VH10, VH11, VH12, VH13, VH14, VH15, VH16, VH17, VH18, VH19, VH20, VH21, VH22, VH23, VH24, VH25, VH26, VH27, VH28, VH29, VH30, VH31 및 VH32들로 이루어지는 그룹으로부터 선택되는 항체 중쇄 가변영역 및/또는 VL1, VL2, VL3, VL4, VL5, VL6, VL7, VL8, VL9, VL10, VL11, VL12, VL13, VL14, VL15, VL16, VL17, VL18, VL19, VL20, VL21, VL22, VL23, VL24, VL25, VL26, VL27, VL28, VL29, VL30, VL31, VL32, VL33 및 VL34들로 이루어지는 그룹으로부터 선택되는 항체 경쇄 가변영역 및 면역학적으로 기능적인 단편들, 유도체들, 이들 경쇄 및 중쇄 가변영역들의 돌연변이 단백질(muteins)들 및 변종들을 포함하는 항원 결합 단백질들이 또한 제공된다.
상기 여러 중쇄 및 경쇄 가변영역들 각각의 시퀀스 정렬들을 도 1a 및 도 1b들에 제공한다.
이러한 형태의 항원 결합 단백질들은 대체로 식 "VHx/VLy"로 표기될 수 있으며, 여기에서 "x"는 표 2에 개시된 바와 같은 중쇄 가변영역들의 수에 대응하고 그리고 "y"는 경쇄 가변영역들의 수에 대응한다(대체로, x 및 y는 각각 1 또는 2이다).
[표 2-1]
[표 2-2]
[표 2-3]
[표 2-4]
[표 2-5]
표 2에 개시된 상기 중쇄 가변영역들 각각은 표 2에 나타낸 상기 경쇄 가변영역들 중의 임의의 것과 결합되어 항원 결합 단백질을 형성할 수 있다. 이러한 조합들의 예들에는 VL1, VL2, VL3, VL4, VL5, VL6, VL7, VL8, VL9, VL10, VL11, VL12, VL13, VL14, VL15, VL16, VL17, VL18, VL19, VL20, VL21, VL22, VL23, VL24, VL25, VL26, VL27, VL28, VL29, VL30, VL31, VL32, VL33 또는 VL34들 중의 어느 것과 결합된 VH1; VL1, VL2, VL3, VL4, VL5, VL6, VL7, VL8, VL9, VL10, VL11, VL12, VL13, VL14, VL15, VL16, VL17, VL18, VL19, VL20, VL21, VL22, VL23, VL24, VL25, VL26, VL27, VL28, VL29 또는 VL30들 중의 어느 것과 결합된 VH2; 또는 VL1, VL2, VL3, VL4, VL5, VL6, VL7, VL8, VL9, VL10, VL11, VL12, VL13, VL14, VL15, VL16, VL17, VL18, VL19, VL20, VL21, VL22, VL23, VL24, VL25, VL26, VL27, VL28, VL29, VL30, VL31, VL32, VL33 또는 VL34들 중의 어느 것과 결합된 VH3 등이 포함된다.
일부 경우들에 있어서, 상기 항원 결합 단백질은 표 2에 개시된 것들로부터의 적어도 하나의 중쇄 가변영역 및/또는 하나의 경쇄 가변영역을 포함한다. 일부 경우들에 있어서, 상기 항원 결합 단백질은 표 2에 개시된 것들로부터의 적어도 2개의 서로 다른 중쇄 가변영역들 및/또는 경쇄 가변영역들을 포함한다. 이러한 항원 결합 단백질의 구체예는 (a) 하나의 VH1 및 (b) VH2, VH3, VH4, VH5, VH6, VH7, VH8, VH9, VH10, VH11, VH12, VH13, VH14, VH15, VH16, VH17, VH18, VH19, VH20, VH21, VH22, VH23, VH24, VH25, VH26, VH27, VH28, VH29, VH30, VH31 또는 VH32들 중의 하나를 포함한다. 다른 구체예는 (a) 하나의 VH2 및 (b) VH1, VH3, VH4, VH5, VH6, VH7, VH8, VH9, VH10, VH11, VH12, VH13, VH14, VH15, VH16, VH17, VH18, VH19, VH20, VH21, VH22, VH23, VH24, VH25, VH26, VH27, VH28, VH29, VH30, VH31 또는 VH32들 중의 하나를 포함한다. 또 다른 구체예는 (a) 하나의 VH3 및 (b) VH1, VH2, VH4, VH5, VH6, VH7, VH8, VH9, VH10, VH11, VH12, VH13, VH14, VH15, VH16, VH17, VH18, VH19, VH20, VH21, VH22, VH23, VH24, VH25, VH26, VH27, VH28, VH29, VH30, VH31 또는 VH32들 중의 하나를 포함한다.
다시 이러한 항원 결합 단백질의 다른 구체예는 (a) 하나의 VL1 및 (b) VL2, VL3, VL4, VL5, VL6, VL7, VL8, VL9, VL10, VL11, VL12, VL13, VL14, VL15, VL16, VL17, VL18, VL19, VL20, VL21, VL22, VL23, VL24, VL25, VL26, VL27, VL28, VL29, VL30, VL31, VL32, VL33 또는 VL34들 중의 하나를 포함한다. 다시 이러한 항원 결합 단백질의 다른 구체예는 (a) 하나의 VL2 및 (b) VL1, VL3, VL4, VL5, VL6, VL7, VL8, VL9, VL10, VL11, VL12, VL13, VL14, VL15, VL16, VL17, VL18, VL19, VL20, VL21, VL22, VL23, VL24, VL25, VL26, VL27, VL28, VL29, VL30, VL31, VL32, VL33 및 VL34들 중의 하나를 포함한다. 다시 이러한 항원 결합 단백질의 다른 구체예는 (a) 하나의 VL3 및 (b) VL1, VL2, VL4, VL5, VL6, VL7, VL8, VL9, VL10, VL11, VL12, VL13, VL14, VL15, VL16, VL17, VL18, VL19, VL20, VL21, VL22, VL23, VL24, VL25, VL26, VL27, VL28, VL29, VL30, VL31, VL32, VL33 또는 VL34들 중의 하나를 포함한다.
중쇄 가변영역들의 상기 여러 조합들은 경쇄 가변영역들의 여러 조합들 중의 임의의 것과 결합될 수 있다.
다른 경우들에 있어서, 상기 항원 결합 단백질은 2개의 동일한 경쇄 가변영역들 및/또는 2개의 동일한 중쇄 가변영역들을 포함한다. 구체예로서, 상기 항원 결합 단백질은 표 2에 개시된 바와 같은 경쇄 가변영역들의 쌍들과 중쇄 가변영역들의 쌍들의 조합들의 2개의 경쇄 가변영역들 및 2개의 중쇄 가변영역들을 포함하는 항체 또는 면역학적으로 기능적인 단편이 될 수 있다.
제공되는 일부 항원 결합 단백질들은 단지 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14 또는 15개의 아미노산 잔기들에서의 VH1, VH2, VH3, VH4, VH5, VH6, VH7, VH8, VH9, VH10, VH11, VH12, VH13, VH14, VH15, VH16, VH17, VH18, VH19, VH20, VH21, VH22, VH23, VH24, VH25, VH26, VH27, VH28, VH29, VH30, VH31 및 VH32들로부터 선택되는 중쇄 가변 도메인의 시퀀스와는 다른 아미노산들의 시퀀스를 포함하는 중쇄 가변 도메인을 포함하며, 여기에서 이러한 시퀀스 각각의 차이는 앞서의 가변영역 시퀀스들에 대해 15개 이하의 아미노산 변화들의 결과를 가져오는 탈락들, 삽입들 및/또는 치환들을 갖는 하나의 아미노산의 독립적인 탈락, 삽입 또는 치환이다. 일부 항원 결합 단백질들에서의 상기 중쇄 가변영역은 VH1, VH2, VH3, VH4, VH5, VH6, VH7, VH8, VH9, VH10, VH11, VH12, VH13, VH14, VH15, VH16, VH17, VH18, VH19, VH20, VH21, VH22, VH23, VH24, VH25, VH26, VH27, VH28, VH29, VH30, VH31 및 VH32의 상기 중쇄 가변영역의 아미노산 시퀀스들에 대해 적어도 70%, 75%, 80%, 85%, 90%, 95%, 97% 또는 99%의 시퀀스 동일성을 갖는 아미노산의 시퀀스를 포함한다.
특정의 항원 결합 단백질들은 단지 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14 또는 15개의 아미노산 잔기들에서의 VL1, VL2, VL3, VL4, VL5, VL6, VL7, VL8, VL9, VL10, VL11, VL12, VL13, VL14, VL15, VL16, VL17, VL18, VL19, VL20, VL21, VL22, VL23, VL24, VL25, VL26, VL27, VL28, VL29, VL30, VL31, VL32, VL33 또는 VL34들로부터 선택되는 경쇄 가변 도메인의 시퀀스와는 다른 아미노산들의 시퀀스를 포함하는 경쇄 가변 도메인을 포함하며, 여기에서 이러한 시퀀스 각각의 차이는 앞서의 가변 도메인 시퀀스들에 대해 15개 이하의 아미노산 변화들의 결과를 가져오는 탈락들, 삽입들 및/또는 치환들을 갖는 하나의 아미노산의 독립적인 탈락, 삽입 또는 치환이다. 일부 항원 결합 단백질들에서의 상기 경쇄 가변영역은 VL1, VL2, VL3, VL4, VL5, VL6, VL7, VL8, VL9, VL10, VL11, VL12, VL13, VL14, VL15, VL16, VL17, VL18, VL19, VL20, VL21, VL22, VL23, VL24, VL25, VL26, VL27, VL28, VL29, VL30, VL31, VL32, VL33 또는 VL34들의 상기 경쇄 가변영역의 아미노산 시퀀스들에 대해 적어도 70%, 75%, 80%, 85%, 90%, 95%, 97% 또는 99%의 시퀀스 동일성을 갖는 아미노산의 시퀀스를 포함한다.
또 다른 항원 결합 단백질들, 예를 들면, 항체들 또는 면역학적으로 기능적인 단편들은 기재된 바와 같은 변종의 중쇄 및 변종의 경쇄의 변종의 형태들을 포함한다.
CDRs
본 명세서에서 기술되는 상기 항원 결합 단백질들은 그 안으로 하나 또는 그 이상의 CDRs들이 분지되거나, 삽입되거나 및/또는 결합되는 폴리펩티드들이다. 항원 결합 단백질은 1, 2, 3, 4, 5 또는 6개의 CDRs들을 가질 수 있다. 따라서 항원 결합 단백질은, 예를 들면, 하나의 중쇄 CDR1("CDRHl") 및/또는 하나의 중쇄 CDR2("CDRH2") 및/또는 하나의 중쇄 CDR3("CDRH3") 및/또는 하나의 경쇄 CDRl("CDRLl") 및/또는 하나의 경쇄 CDR2("CDRL2") 및/또는 하나의 경쇄 CDR3("CDRL3")들을 가질 수 있다. 일부 항원 결합 단백질들에는 CDRH3 및 CDRL3 둘 다 포함된다. 특정의 중쇄 및 경쇄 CDRs들은 각각 표 3a(예시적인 CDRH 시퀀스들) 및 표 3b(예시적인 CDRL 시퀀스들)에서 확인된다.
주어진 항체의 상보적 결합 영역들(CDRs) 및 골격영역들(FR)은 카바트와 그의 동료들(Kabat et al.)의 in Sequences of Proteins of Immunological Interest, 5th Ed., US Dept. of Health and Human Services, PHS, NIH, NIH Publication no. 91-3242, 1991 문헌에 의해 기술된 시스템을 사용하여 동정될 수 있다. 본 명세서에서 기술되는 특정의 항체들은 표 3a(CDRHs) 및 표 3b(CDRLs)들에 제공된 하나 또는 그 이상의 CDRs들의 아미노산 시퀀스들에 대해 동일하거나 또는 상당한 시퀀스 동일성을 갖는 하나 또는 그 이상의 아미노산 시퀀스들을 포함한다.
[표 3a-1]
[표 3a-2]
[표 3b-1]
[표 3b-2]
[표 3b-3]
자연적으로 발생하는 항체 내의 CDRs들의 구조 및 특성들은 위에서 기술하였다. 간단하게, 전통적인 항체에 있어서, 상기 CDRs들은 항원 결합 및 인식에 대해 책임이 있는 영역들을 구성하는 상기 중쇄 및 경쇄 가변영역 내의 골격 내에 매립된다. 가변영역은 골격영역(카바트와 그의 동료들의 위의 1991 문헌에 의해 골격영역 1-4, FRl, FR2, FR3 및 FR4들로 표기된; 또한 초티아와 레스크의 위의 1987 문헌을 참조하시오) 내에 적어도 3개의 중쇄 또는 경쇄 CDRs들(Kabat et al, 1991, Sequences of Proteins of Immunological Interest, Public Health Service N.I.H., Bethesda, MD을 참조하시오; 또한 초티아와 레스크(Chothia and Lesk)의 1987, J. MoI. Biol. 196:901-917; 및 초티아와 그의 동료들(Chothia et al)의 1989, Nature 342: 877- 883를 참조하시오)을 포함한다. 그러나 본 명세서에서 제공되는 상기 CDRs들은 단지 전통적인 항체 구조의 상기 항원 결합 도메인을 한정하는 데 사용될 뿐만 아니라 본 명세서에서 기술되는 바와 같이 다양한 다른 폴리펩티드 구조들 내에 매립될 수도 있다.
하나의 관점에 있어서, 제공되는 상기 CDRs들은 (a) (i) SEQ ID NO: 136-147들로 이루어지는 그룹으로부터 선택되는 CDRHl; (ii) SEQ ID NO: 148-164들로 이루어지는 그룹으로부터 선택되는 CDRH2; (iii) SEQ ID NO: 165-190들로 이루어지는 그룹으로부터 선택되는 CDRH3; 및 (iv) 5, 4, 3, 2 또는 1의 아미노산들의 하나 또는 그 이상의 아미노산 치환들, 탈락들 또는 삽입들을 포함하는 (i), (ii) 및 (iii)의 CDRH;들로 이루어지는 그룹으로부터 선택되는 CDRH; (B) (i) SEQ ED NO: 191-210들로 이루어지는 그룹으로부터 선택되는 CDRLl; (ii) SEQ ID NO: 211-224들로 이루어지는 그룹으로부터 선택되는 CDRL2; (iii) SEQ ID NO: 225-246들로 이루어지는 그룹으로부터 선택되는 CDRL3; 및 (iv) 5, 4, 3, 2 또는 1의 아미노산들의 하나 또는 그 이상의 아미노산 치환들, 탈락들 또는 삽입들을 포함하는 (i), (ii) 및 (iii)의 CDRH;들로 이루어지는 그룹으로부터 선택되는 CDRL;들로 이루어지는 그룹으로부터 선택되는 CDRL;들이다.
다른 관점에 있어서, 항원 결합 단백질은 표 3a 및 표 3b들에 기재된 상기 CDRs들의 1, 2, 3, 4, 5 또는 6의 변종 형태들을 포함하며, 각각은 표 3a 및 표 3b들에 기재된 CDR 시퀀스에 대하여 적어도 80%, 85%, 90% 또는 95%의 시퀀스 동일성을 갖는다. 일부 항원 결합 단백질들은 표 3a 및 표 3b들에 기재된 상기 CDRs들의 1, 2, 3, 4, 5 또는 6들을 포함하며, 각각은 이들 표들 내에 기재된 상기 CDRs들로부터 1, 2, 3, 4 또는 5개 이하로 다르다.
또 다른 관점에 있어서, 본 명세서에서 기술된 상기 CDRs들은 연관된 모노클로날 항체들의 그룹들로부터 유도되는 합치 시퀀스들을 포함한다. 본 명세서에서 기술하는 바와 같이, "합치 시퀀스"는 주어진 아미노산 시퀀스들 내에서 변하는 다수의 시퀀스들 및 가변 아미노산들 중에서 공통인 보존된 아미노산들을 갖는 아미노산 시퀀스들을 의미한다. 제공되는 상기 CDR 합치 시퀀스들은 CDRHl, CDRH2, CDRH3, CDRLl, CDRL2 및 CDRL3의 각각에 대응하는 CDRs들을 포함한다.
합치 시퀀스들은 항-c-fms 항체들의 VH 및 VL에 대응하는 상기 CDRs들의 표준 계통발생 분석(standard phylogenic analyses)들을 사용하여 결정된다. 상기 합치 시퀀스들은 VH 및 VL에 대응하는 동일한 시퀀스 내의 인접적인 상기 CDRs들을 보존하는 것에 의해 결정된다. 간단하게, 비교 정렬들 및 계통발생의 추론을 용이하게 하기 위하여 VH 및 VL 중의 어느 하나의 전체 가변 도메인들에 대응하는 아미노산 시퀀스들을 FASTA 포맷팅(formatting)으로 전환시켰다. 다음으로, 이들 시퀀스들의 골격영역들을 인공의 링커 시퀀스("GGGAAAGGGAAA" (SEQ ID NO:325))로 대체시켜 동시발생의 사건(예를 들면, 생식계열 골격 유산을 우연히 공유하는 비연관된 항체들 등과 같은)들로 인한 임의의 아미노산 위치 가중 바이어스를 도입함이 없이 상기 CDRs들 단독의 검사가 수행되도록 하는 한편으로, VH 또는 VL에 대응하는 동일한 시퀀스 내에서의 인접적인 CDRs들을 여전히 유지하도록 한다. 계속해서 이러한 포맷(format)의 VH 또는 VL 시퀀스들을 표준 ClutalW-형 알고리즘을 사용하는 프로그램을 이용하여 시퀀스 유사성 정렬 해석에 적용시켰다(톰슨과 그의 동료들(Thompson et al)의 1994, Nucleic Acids Res. 22:4673- 4680)를 참조하시오). 2.0의 갭 연장 페널티와 함께 8.0의 갭 생성 페널티를 사용하였다. 이 프로그램은 가지 길이 비교 및 그룹화를 통하여 시퀀스 그룹들의 유사성 및 차별성을 구축하고 그리고 설명하기 위하여 UPGMA(unweighted pair group method using arithmetic averages ; 산술평균을 사용하는 비가중된 쌍그룹법) 또는 네이버-결합 방법(Neighbor-Joining methods ; 사이토우와 네이(Saitou and Nei)의 1987, Molecular Biology and Evolution 4:406-425을 참조하시오)들 중 어느 하나를 사용하는 시퀀스 유사성 정렬들에 기초하여 유사하게 계통발생도(phylograms ; 계통수 도식)들을 생성하였다. 두 방법들은 유사한 결과들을 생성하였으나, 보다 단순하고 그리고 보다 많은 가정들의 보수적인 설정을 사용하는 방법이기 때문에 UPGMA-유도 계통수들이 궁극적으로 사용되었다. UPGMA-유도 계통수들을 도 2에 나타내었으며, 여기에서 시퀀스들의 유사한 그룹들은 그룹 내의 개개 시퀀스들 중의 100개의 잔기들(규모에 대한 계통수 도식들에서의 범례) 당 15개 이하의 치환들을 갖는 것으로 한정되었으며, 합치 시퀀스 수집들을 한정하는 데 사용되었다.
도 2에 도시된 바와 같이, 본 명세서에서 제공되는 다양한 상기 항원 결합 단백질들의 혈통 분석은 그룹 A, B 및 C들로 표기된 밀접하게 연관된 계통발생적인 클론들의 세 그룹들의 결과를 가져왔다.
그룹 A의 다양한 CDR 영역들의 합치 시퀀스들은 다음과 같다:
a. 일반식 GYTX1TSYGIS(SEQ ID NO: 307)의 CDRH1, 여기에서 X1은 F 및 L로 이루어지는 그룹으로부터 선택되는 것임;
b. 일반식 WISAYNGNX1NYAQKX2QG(SEQ ID NO: 308)의 CDRH2, 여기에서 X1은 T 및 P로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X2는 L 및 F로 이루어지는 그룹으로부터 선택되는 것임;
c. 일반식 X1X2X3X4X4X5FGEX6X7X8X9FDY(SEQ ID NO: 309)의 CDRH3, 여기에서 X1은 E 및 D로 이루어지는 그룹으로부터 선택되는 것이고, X2는 S 및 Q로 이루어지는 그룹으로부터 선택되는 것이고, X3은 G 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X4는 L 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X5는 W 및 G로 이루어지는 그룹으로부터 선택되는 것이고, X6은 V 및 L로 이루어지는 그룹으로부터 선택되는 것이고, X7은 E 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X8은 G 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X9는 F 및 L로 이루어지는 그룹으로부터 선택되는 것임;
d. 일반식 KSSX1GVLX2SSX3NKNX4LA(SEQ ID NO: 310)의 CDRL1, 여기에서 X1은 Q 및 S로 이루어지는 그룹으로부터 선택되는 것이고, X2는 D 및 Y로 이루어지는 그룹으로부터 선택되는 것이고, X3은 N 및 D로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X4는 F 및 Y로 이루어지는 그룹으로부터 선택되는 것임;
e. 일반식 WASX1RES(SEQ ID NO: 311)의 CDRL2, 여기에서 X1은 N 및 T로 이루어지는 그룹으로부터 선택되는 것임;
f. 일반식 QQYYX1X2PX3T(SEQ ID NO:312)의 CDRL3, 여기에서 X1은 S 및 T로 이루어지는 그룹으로부터 선택되는 것이고, X2는 D 및 T로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X3은 F 및 P로 이루어지는 그룹으로부터 선택되는 것임.
그룹 B의 다양한 CDR 영역들의 합치 시퀀스들은 다음과 같다:
a. 일반식 GFTX1X2X3AWMS(SEQ ID NO: 313)을 갖는 CDRHl, 여기에서 X1은 F 및 V로 이루어지는 그룹으로부터 선택되는 것이고, X2는 S 및 N으로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X3은 N 및 T로 이루어지는 그룹으로부터 선택되는 것임;
b. 일반식 RIKX1KTDGX2TX3DX4AAPVKG(SEQ ID NO: 314)을 갖는 CDRH2, 여기에서 X1은 S 및 T로 이루어지는 그룹으로부터 선택되는 것이고, X2는 G 및 W로 이루어지는 그룹으로부터 선택되는 것이고, X3은 T 및 A로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X4는 Y 및 N으로 이루어지는 그룹으로부터 선택되는 것임;
c. 일반식 X1X2X3X4X5X6X7X8X9X10X11X12X13YYGX14DV(SEQ ID NO: 315)을 갖는 CDRH3, 여기에서 X1은 E, D 및 G로 이루어지는 그룹으로부터 선택되는 것이고, X2는 Y, L 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X3은 Y, R, G 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X4는 H, G, S 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X5는 I, A, L 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X6은 L, V, T, P 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X7은 T, V, Y, G, W 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X8은 G, V, S 및 T로 이루어지는 그룹으로부터 선택되는 것이고, X9는 S, T, D, N 및 G로 이루어지는 그룹으로부터 선택되는 것이고, X10은 G, F, P 및 Y로 이루어지는 그룹으로부터 선택되는 것이고, X11은 G, Y 및 N으로 이루어지는 그룹으로부터 선택되는 것이고, X12는 V 및 Y로 이루어지는 그룹으로부터 선택되는 것이고, X13은 W, S 및 Y로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X14는 M, T 및 V로 이루어지는 그룹으로부터 선택되는 것임;
d. 일반식 QASQDIX1NYLN(SEQ ID NO: 316)을 갖는 CDRLl, 여기에서 X1은 S 및 N으로 이루어지는 그룹으로부터 선택되는 것임;
e. 일반식 DX1SNLEX2(SEQ ID NO: 317)을 갖는 CDRL2, 여기에서 X1은 A 및 T로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X2는 T 및 P로 이루어지는 그룹으로부터 선택되는 것임; 그리고
f. 일반식 QQYDX1LX2T(SEQ ID NO: 318)을 갖는 CDRL3, 여기에서 X1은 N 및 D로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X2는 L 및 I로 이루어지는 그룹으로부터 선택되는 것임.
그룹 C의 다양한 CDR 영역들의 합치 시퀀스들은 다음과 같다:
a. 일반식 GFTFX1SYGMH(SEQ ID NO: 319)을 갖는 CDRHl, 여기에서 X1은 S 및 I로 이루어지는 그룹으로부터 선택되는 것임;
b. 일반식 VIWYDGSNX1YYADSVKG(SEQ ID NO: 320)을 갖는 CDRH2, 여기에서 X1은 E 및 K로 이루어지는 그룹으로부터 선택되는 것임;
c. 일반식 SSX1X2X3YX4MDV(SEQ ID NO: 321)을 갖는 CDRH3, 여기에서 X1은 G, S 및 W로 이루어지는 그룹으로부터 선택되는 것이고, X2는 N, D 및 S로 이루어지는 그룹으로부터 선택되는 것이고, X3은 Y 및 F로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X4는 D 및 G로 이루어지는 그룹으로부터 선택되는 것임;
d. 일반식 QASX1DIX2NX3LN(SEQ ID NO: 322)을 갖는 CDRLl, 여기에서 X1은 Q 및 H로 이루어지는 그룹으로부터 선택되는 것이고, X2는 S 및 N으로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X3은 F 및 Y로 이루어지는 그룹으로부터 선택되는 것임;
e. 일반식 DASNLEX1(SEQ ID NO: 323)을 갖는 CDRL2, 여기에서 X1은 T 및 I로 이루어지는 그룹으로부터 선택되는 것임; 그리고
f. 일반식 QX1YDX2X3PX4T(SEQ ID NO: 324)를 갖는 CDRL3, 여기에서 X1은 Q 및 R로 이루어지는 그룹으로부터 선택되는 것이고, X2는 N 및 D로 이루어지는 그룹으로부터 선택되는 것이고, X3은 L 및 F로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X4는 F, L 및 I로 이루어지는 그룹으로부터 선택되는 것임.
일부 경우들에 있어서, 상기 항원 결합 단백질은 상기 합치 시퀀스들 중의 하나를 갖는 적어도 하나의 CDRHl, CDRH2 또는 CDRH3를 포함한다. 일부 경우들에 있어서, 상기 항원 결합 단백질은 상기 합치 시퀀스들 중의 하나를 갖는 적어도 하나의 CDRLl, CDRL2 또는 CDRL3를 포함한다. 다른 경우들에 있어서, 상기 항원 결합 단백질은 상기 합치 시퀀스들에 따른 적어도 2개의 CDRHs들 및/또는 상기 합치 시퀀스들에 따른 적어도 2개의 CDRLs들을 포함한다. 하나의 관점에 있어서, 상기 CDRHs 및/또는 CDRLs들은 서로 다른 그룹 A, B 및 C들로부터 유도된다. 다른 경우들에 있어서, 상기 항원 결합 단백질은 상기 동일한 그룹 A, B 또는 C로부터의 적어도 2개의 CDRHs들 및/또는 상기 동일한 그룹 A, B 또는 C로부터의 적어도 2개의 CDRLs들을 포함한다. 다른 관점들에 있어서, 상기 항원 결합 단백질은 상기 그룹 A, B 또는 C들 중의 동일한 그룹으로부터의 CDRHl, CDRH2 및 CDRH3 및/또는 상기 그룹 A, B 또는 C들 중의 동일한 그룹으로부터의 CDRLl, CDRL2 및 CDRL3들을 포함한다.
따라서, 제공되는 일부 항원 결합 단백질들은 그룹 A 합치 시퀀스들로부터의 상기 CDRs들의 1 , 2, 3, 4, 5 또는 6의 전부를 포함한다. 따라서 예를 들면 특정의 항원 결합 단백질들은 앞서 규정한 상기 그룹 A 합치 시퀀스들로부터의 CDRHl, CDRH2, CDRH3, CDRLl, CDRL2 및 CDRL3들을 포함한다. 제공되는 다른 항원 결합 단백질들은 상기 그룹 B 합치 시퀀스들로부터의 상기 CDRs들의 1, 2, 3, 4, 5 또는 6의 전부를 포함한다. 따라서 예를 들면 특정의 항원 결합 단백질들은 앞서 규정한 상기 그룹 B 합치 시퀀스들로부터의 CDRHl, CDRH2, CDRH3, CDRLl, CDRL2 및 CDRL3들을 포함한다. 제공되는 또 다른 항원 결합 단백질들은 상기 그룹 C 합치 시퀀스들로부터의 상기 CDRs들의 1, 2, 3, 4, 5 또는 6의 전부를 포함한다. 따라서 예를 들면 특정의 항원 결합 단백질들은 앞서 규정한 상기 그룹 A 합치 시퀀스들로부터의 CDRHl, CDRH2, CDRH3, CDRLl, CDRL2 및 CDRL3들을 포함한다.
예시적인 항원 결합 단백질들
하나의 관점에 따르면, (A) (i) SEQ ID NOs: 136-147들로 이루어지는 그룹으로부터 선택되는 CDRH1; (ii) SEQ ID NOs: 148-164들로 이루어지는 그룹으로부터 선택되는 CDRH2; (iii) SEQ ID NOs: 165-190들로 이루어지는 그룹으로부터 선택되는 CDRH3; 및 (iv) 5, 4, 3, 2 또는 1의 아미노산들의 하나 또는 그 이상의 아미노산 치환들, 탈락들 또는 삽입들을 포함하는 (i), (ii) 및 (iii)의 CDRH;들로 이루어지는 그룹으로부터 선택되는 CDRH;들로 이루어지는 그룹으로부터 선택되는 하나 또는 그 이상의 중쇄보족결정영역(CDRHs); (B) (i) SEQ ID NOs: 191-210들로 이루어지는 그룹으로부터 선택되는 CDRLl; (ii) SEQ ID NOs: 211-224들로 이루어지는 그룹으로부터 선택되는 CDRL2; (iii) SEQ ID NOs: 225-246들로 이루어지는 그룹으로부터 선택되는 CDRL3; 및 (iv) 5, 4, 3, 2 또는 1의 아미노산들의 하나 또는 그 이상의 아미노산 치환들, 탈락들 또는 삽입들을 포함하는 (i), (ii) 및 (iii)의 CDRL;들로 이루어지는 그룹으로부터 선택되는 하나 또는 그 이상의 경쇄보족결정영역(CDRLs); 또는 (C) (A)의 하나 또는 그 이상의 중쇄 CDRHs 및 (B)의 하나 또는 그 이상의 경쇄 CDRLs들을 포함하는 c-fms를 결합하는 단리된 항원-결합 단백질이 제공된다.
또 다른 구체예에 있어서, 상기 단리된 항원-결합 단백질은 (A) (i) SEQ ID NO: 136-147들로 이루어지는 그룹으로부터 선택되는 CDRHl; (ii) SEQ ID NO: 148-164들로 이루어지는 그룹으로부터 선택되는 CDRH2; 및 (iii) SEQ ID NO: 165-190들로 이루어지는 그룹으로부터 선택되는 CDRH3;들로 이루어지는 그룹으로부터 선택되는 CDRH; (B) (i) SEQ ID NO: 191-210들로 이루어지는 그룹으로부터 선택되는 CDRLl; (ii) SEQ ID NO: 211-224들로 이루어지는 그룹으로부터 선택되는 CDRL2; 및 (iii) SEQ ID NO: 225-246들로 이루어지는 그룹으로부터 선택되는 CDRL3;들로 이루어지는 그룹으로부터 선택되는 CDRL; 또는 (C) (A)의 하나 또는 그 이상의 중쇄 CDRHs들 및 (B)의 하나 또는 그 이상의 경쇄 CDRLs들;을 포함할 수 있다. 하나의 구체예에 있어서, 상기 단리된 항원-결합 단백질은 (A) SEQ ID NO: 136-147의 CDRHl, SEQ ID NO: 148-164의 CDRH2 및 SEQ ID NO: 165-190의 CDRH3 및 (B) SEQ ID NO: 191-210의 CDRLl, SEQ ID NO.211-224의 CDRL2 및 SEQ ID NO: 225-246의 CDRL3들을 포함할 수 있다.
다른 구체예에 있어서, 상기 가변중쇄(VH)는 SEQ ID NO: 70-101들로 이루어지는 그룹으로부터 선택되는 아미노산 시퀀스에 대하여 적어도 70%, 75%, 80%, 85%, 90%, 95%, 97% 또는 99%의 시퀀스 동일성을 가지거나 및/또는 상기 가변경쇄(VL)는 SEQ ID NO: 102-135들로 이루어지는 그룹으로부터 선택되는 아미노산 시퀀스에 대하여 적어도 70%, 75%, 80%, 85%, 90%, 95%, 97% 또는 99%의 시퀀스 동일성을 갖는다. 다른 구체예에 있어서, 상기 VH는 SEQ ID NO: 70-101들로 이루어지는 그룹으로부터 선택되거나 및/또는 상기 VL은 SEQ BD NO: 102-135들로 이루어지는 그룹으로부터 선택된다.
다른 관점에 있어서, 상기 c-fms 서브도메인들인 c-fms의 Ig-형 l-1 및 Ig-형 1-2들을 포함하는 에피토프에 특이적으로 결합하는 단리된 항원 결합 단백질이 또한 제공된다.
또 다른 관점에 있어서, c-fms에 결합하는 단리된 항원-결합 단백질이 제공되며, 상기 항원-결합 단백질은 (1) SEQ ID NOs: 165-190들로 이루어지는 그룹으로부터 선택되는 CDRH3, (2) (i) 2개 이하의 아미노산들의 아미노산 첨가, 탈락 또는 치환에 의한 상기 CDRH3로부터의 아미노산 시퀀스와는 다른 CDRH3; 및 (3) (a) X1X2X3X4X4X5FGEX6X7X8X9FDY(SEQ ID NO: 309), 여기에서 X1은 E 및 D로 이루어지는 그룹으로부터 선택되는 것이고, X2는 S 및 Q로 이루어지는 그룹으로부터 선택되는 것이고, X3은 G 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X4는 L 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X5는 W 및 G로 이루어지는 그룹으로부터 선택되는 것이고, X6은 V 및 L로 이루어지는 그룹으로부터 선택되는 것이고, X7은 E 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X8은 G 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X9는 F 및 L로 이루어지는 그룹으로부터 선택되는 것이고(앞서 기술된 계통발생적인 그룹 A로부터 유도되는 CDRH3 합치 시퀀스); (b) X1X2X3X4X5X6X7X8X9X10X11X12X13YYGX14DV(SEQ ID NO: 315), 여기에서 X1은 E, D 및 G로 이루어지는 그룹으로부터 선택되는 것이고, X2는 Y, L 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X3은 Y, R, G 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X4는 H, G, S 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X5는 I, A, L 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X6은 L, V, T, P 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X7은 T, V, Y, G, W 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X8은 G, V, S 및 T로 이루어지는 그룹으로부터 선택되는 것이고, X9는 S, T, D, N 및 G로 이루어지는 그룹으로부터 선택되는 것이고, X10은 G, F, P 및 Y로 이루어지는 그룹으로부터 선택되는 것이고, X11은 G, Y 및 N으로 이루어지는 그룹으로부터 선택되는 것이고, X12는 V 및 Y로 이루어지는 그룹으로부터 선택되는 것이고, X13은 W, S 및 Y로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X14는 M, T 및 V로 이루어지는 그룹으로부터 선택되는 것임(앞서 기술된 계통발생적인 그룹 B로부터 유도되는 CDRH3 합치 시퀀스); 및 (c) SSX1X2X3YX4MDV(SEQ ID NO: 321), 여기에서 X1은 G, S 및 W로 이루어지는 그룹으로부터 선택되는 것이고, X2는 N, D 및 S로 이루어지는 그룹으로부터 선택되는 것이고, X3은 Y 및 F로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X4는 D 및 G로 이루어지는 그룹으로부터 선택되는 것이고(앞서 기술된 계통발생적인 그룹 C로부터 유도되는 CDRH3 합치 시퀀스);들로 이루어지는 그룹으로부터 선택되는 CDRH3 아미노산 시퀀스; 또는 (B) (1) SEQ ID NOs: 225-246들로 이루어지는 그룹으로부터 선택되는 CDRL3, (2) (i) 2개 이하의 아미노산들의 아미노산 첨가, 탈락 또는 치환에 의한 상기 CDRL3로부터의 아미노산 시퀀스와는 다른 CDRL3; 및 (3) (a) QQYYX1X2PX3T(SEQ ID NO:312), 여기에서 X1은 S 및 T로 이루어지는 그룹으로부터 선택되는 것이고, X2는 D 및 T로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X3은 F 및 P로 이루어지는 그룹으로부터 선택되는 것이고(앞서 기술된 계통발생적인 그룹 A로부터 유도되는 CDRL3 합치 시퀀스); (b) QQYDX1LX2T(SEQ ID NO: 318), 여기에서 X1은 N 및 D로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X2는 L 및 I로 이루어지는 그룹으로부터 선택되는 것임(앞서 기술된 계통발생적인 그룹 B로부터 유도되는 CDRL3 합치 시퀀스); 및 (c) QX1YDX2X3PX4T(SEQ ID NO: 324), 여기에서 X1은 Q 및 R로 이루어지는 그룹으로부터 선택되는 것이고, X2는 N 및 D로 이루어지는 그룹으로부터 선택되는 것이고, X3은 L 및 F로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X4는 F, L 및 I로 이루어지는 그룹으로부터 선택되는 것임(앞서 기술된 계통발생적인 그룹 C로부터 유도되는 CDRL3 합치 시퀀스);들로 이루어지는 그룹으로부터 선택되는 CDRL3 아미노산 시퀀스;들로 이루어지는 그룹으로부터 선택되는 경쇄 상보적 결합 영역(CDRL);들을 포함한다.
하나의 구체예에 있어서, c-fms를 결합하는 상기 항원 결합 단백질은 그룹 A, B 또는 C의 합치 시퀀스에 따른 CDRH3 및/또는 그룹 A, B 또는 C의 합치 시퀀스에 따른 CDRL3 및 상기 그룹들 중의 임의의 것의 CDRH1 및/또는 CDRH2 및/또는 상기 그룹들 중의 임의의 것의 CDRLl 및/또는 CDRL2들을 포함한다.
하나의 구체예에 있어서, c-fms를 결합하는 상기 단리된 항원 결합 단백질은 위의 그룹 A의 CDRH3 및/또는 CDRL3 및 하기의 것들로 이루어지는 그룹으로부터 선택되는 CDR들을 포함함:
(1) (a) SEQ ID NOs: 136-147의 CDRH1; (b) 아미노산 시퀀스에서 2개 이하의 아미노산들의 아미노산 첨가, 탈락 또는 치환에 의한 (a)의 상기 CDRH1과는 CDRH1; 및 (c) GYTX1TSYGIS(SEQ ID NO: 307)들로 이루어지는 그룹으로부터 선택되는 CDRH1 아미노산 시퀀스, 여기에서 X1은 F 및 L로 이루어지는 그룹으로부터 선택되는 것임;들로 이루어지는 그룹으로부터 선택되는 CDRHl;
(2) (a) SEQ ID NOs: 148-164의 CDRH2; (b) 아미노산 시퀀스에서 2개 이하의 아미노산들의 아미노산 첨가, 탈락 또는 치환에 의한 (a)의 상기 CDRH2와는 다른 CDRH2; 및 (c) WISAYNGNX1NYAQKX2QG(SEQ ID NO: 308)들로 이루어지는 그룹으로부터 선택되는 CDRH2 아미노산 시퀀스, 여기에서 X1은 T 및 P로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X2는 L 및 F로 이루어지는 그룹으로부터 선택되는 것임;들로 이루어지는 그룹으로부터 선택되는 CDRH2;
(3) (a) SEQ BD NOs: 191-210의 CDRL1; (b) 아미노산 시퀀스에서 2개 이하의 아미노산들의 아미노산 첨가, 탈락 또는 치환에 의한 (a)의 상기 CDRL1과는 다른 CDRL1; 및 (c) KSSX1GVLX2SSX3NKNX4LA(SEQ ID NO: 310)들로 이루어지는 그룹으로부터 선택되는 CDRL1 아미노산 시퀀스, 여기에서 X1은 Q 및 S로 이루어지는 그룹으로부터 선택되는 것이고, X2는 D 및 Y로 이루어지는 그룹으로부터 선택되는 것이고, X3은 N 및 D로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X4는 F 및 Y로 이루어지는 그룹으로부터 선택되는 것임;들로 이루어지는 그룹으로부터 선택되는 CDRL1; 및
(4) (a) SEQ ID NOs: 211-224의 CDRL2; (b) 아미노산 시퀀스에서 2개 이하의 아미노산들의 아미노산 첨가, 탈락 또는 치환에 의한 (a)의 상기 CDRL2과는 다른 CDRL2; 및 (c) WASX1RES(SEQ ID NO: 311)들로 이루어지는 그룹으로부터 선택되는 CDRL2 아미노산 시퀀스, 여기에서 X1은 N 및 T로 이루어지는 그룹으로부터 선택되는 것임;들로 이루어지는 그룹으로부터 선택되는 CDRL2.
하나의 구체예에 있어서, c-fms를 결합하는 상기 단리된 항원 결합 단백질은 위의 그룹 B의 CDRH3 및/또는 CDRL3 및 하기의 것들로 이루어지는 그룹으로부터 선택되는 CDR들을 포함함:
(1) (a) SEQ ID NOs: 136-147의 CDRH1; (b) 아미노산 시퀀스에서 2개 이하의 아미노산들의 아미노산 첨가, 탈락 또는 치환에 의한 (a)의 상기 CDRH1과는 CDRH1; 및 (c) GFTX1X2X3AWMS(SEQ ID NO: 313)들로 이루어지는 그룹으로부터 선택되는 CDRHl 아미노산 시퀀스, 여기에서 X1은 F 및 V로 이루어지는 그룹으로부터 선택되는 것이고, X2는 S 및 N으로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X3은 N 및 T로 이루어지는 그룹으로부터 선택되는 것임;들로 이루어지는 그룹으로부터 선택되는 CDRH1;
(2) (a) SEQ ID NOs: 148-164의 CDRH2; (b) 아미노산 시퀀스에서 2개 이하의 아미노산들의 아미노산 첨가, 탈락 또는 치환에 의한 (a)의 상기 CDRH2와는 다른 CDRH2; 및 (c) RIKX1KTDGX2TX3DX4AAPVKG(SEQ ID NO: 314)들로 이루어지는 그룹으로부터 선택되는 CDRH2 아미노산 시퀀스, 여기에서 X1은 S 및 T로 이루어지는 그룹으로부터 선택되는 것이고, X2는 G 및 W로 이루어지는 그룹으로부터 선택되는 것이고, X3은 T 및 A로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X4는 Y 및 N으로 이루어지는 그룹으로부터 선택되는 것임;들로 이루어지는 그룹으로부터 선택되는 CDRH2;
(3) (a) SEQ BD NOs: 191-210의 CDRL1; (b) 아미노산 시퀀스에서 2개 이하의 아미노산들의 아미노산 첨가, 탈락 또는 치환에 의한 (a)의 상기 CDRL1과는 다른 CDRL1; 및 (c) QASQDIX1NYLN(SEQ ID NO: 316)들로 이루어지는 그룹으로부터 선택되는 CDRLl 아미노산 시퀀스, 여기에서 X1은 S 및 N으로 이루어지는 그룹으로부터 선택되는 것임;들로 이루어지는 그룹으로부터 선택되는 CDRL1; 및
(4) (a) SEQ ID NOs: 211-224의 CDRL2; (b) 아미노산 시퀀스에서 2개 이하의 아미노산들의 아미노산 첨가, 탈락 또는 치환에 의한 (a)의 상기 CDRL2과는 다른 CDRL2; 및 (c) DX1SNLEX2(SEQ ID NO: 317)들로 이루어지는 그룹으로부터 선택되는 CDRL2 아미노산 시퀀스, 여기에서 X1은 A 및 T로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X2는 T 및 P로 이루어지는 그룹으로부터 선택되는 것임;들로 이루어지는 그룹으로부터 선택되는 CDRL2.
하나의 구체예에 있어서, c-fms를 결합하는 상기 단리된 항원 결합 단백질은 위의 그룹 C의 CDRH3 및 CDRL3 및 하기의 것들로 이루어지는 그룹으로부터 선택되는 CDR들을 포함함:
(1) (a) SEQ ID NOs: 136-147의 CDRH1; (b) 아미노산 시퀀스에서 2개 이하의 아미노산들의 아미노산 첨가, 탈락 또는 치환에 의한 (a)의 상기 CDRH1과는 CDRH1; 및 (c) GFTFX1SYGMH(SEQ ID NO: 319)들로 이루어지는 그룹으로부터 선택되는 CDRHl 아미노산 시퀀스, 여기에서 X1은 S 및 I로 이루어지는 그룹으로부터 선택되는 것임;들로 이루어지는 그룹으로부터 선택되는 CDRH1;
(2) (a) SEQ ID NOs: 148-164의 CDRH2; (b) 아미노산 시퀀스에서 2개 이하의 아미노산들의 아미노산 첨가, 탈락 또는 치환에 의한 (a)의 상기 CDRH2와는 다른 CDRH2; 및 (c) VIWYDGSNX1YYADSVKG(SEQ ID NO: 320)들로 이루어지는 그룹으로부터 선택되는 CDRH2 아미노산 시퀀스, 여기에서 X1은 E 및 K로 이루어지는 그룹으로부터 선택되는 것임;들로 이루어지는 그룹으로부터 선택되는 CDRH2;
(3) (a) SEQ BD NOs: 191-210의 CDRL1; (b) 아미노산 시퀀스에서 2개 이하의 아미노산들의 아미노산 첨가, 탈락 또는 치환에 의한 (a)의 상기 CDRL1과는 다른 CDRL1; 및 (c) QASX1DIX2NX3LN(SEQ ID NO: 322)들로 이루어지는 그룹으로부터 선택되는 CDRLl 아미노산 시퀀스, 여기에서 X1은 Q 및 H로 이루어지는 그룹으로부터 선택되는 것이고, X2는 S 및 N으로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X3은 F 및 Y로 이루어지는 그룹으로부터 선택되는 것임;들로 이루어지는 그룹으로부터 선택되는 CDRL1;
(4) (a) SEQ ID NOs: 211-224의 CDRL2; (b) 아미노산 시퀀스에서 2개 이하의 아미노산들의 아미노산 첨가, 탈락 또는 치환에 의한 (a)의 상기 CDRL2과는 다른 CDRL2; 및 (c) DASNLEX1(SEQ ID NO: 323)들로 이루어지는 그룹으로부터 선택되는 CDRL2 아미노산 시퀀스, 여기에서 X1은 T 및 I로 이루어지는 그룹으로부터 선택되는 것임;들로 이루어지는 그룹으로부터 선택되는 CDRL2.
또 다른 구체예에 있어서, 본 명세서에서 기술되는 상기 단리된 항원 결합 단백질은 상기 CDRH 합치 시퀀스들 중의 적어도 하나를 포함하는 제1아미노산 시퀀스 및 상기 CDRL 합치 시퀀스들 중의 적어도 하나를 포함하는 제2아미노산 시퀀스들을 포함한다. 하나의 관점에 있어서, 상기 제1아미노산 시퀀스는 상기 CDRH 합치 시퀀스들 중의 적어도 2개를 포함하거나 및/또는 상기 제2아미노산 시퀀스는 상기 합치 시퀀스들 중의 적어도 2개를 포함한다. 다른 하나의 관점에 있어서, 상기 제1아미노산 시퀀스는 상기 그룹 A, B 또는 C들 중의 동일한 그룹의 적어도 2개의 CDRHs들을 포함하거나 및/또는 상기 제2아미노산 시퀀스는 상기 그룹 A, B 또는 C들 중의 동일한 그룹의 적어도 2개의 CDRLs들을 포함한다. 또 다른 관점들에 있어서, 상기 제1 및 제2아미노산 시퀀스들은 각각 상기 그룹 A, B 또는 C들 중의 동일한 그룹의 적어도 하나의 CDRH 및 하나의 CDRL을 포함한다. 또 다른 관점에 있어서, 상기 제1아미노산 시퀀스는 상기 그룹 A, B 또는 C들 중의 동일한 그룹의 CDRHl, CDRH2 및 CDRH3를 포함하거나 및/또는 상기 제2아미노산 시퀀스는 상기 그룹 A, B 또는 C들 중의 동일한 그룹의 CDRLl, CDRL2 및 CDRL3를 포함한다.
특정의 구체예들에 있어서, 상기 제1 및 제2아미노산 시퀀스들은 서로에 대하여 공유적으로 결합된다.
또 다른 구체예에 있어서, 상기 단리된 항원-결합 단백질의 상기 제1아미노산 시퀀스는 SEQ ID NO: 165-190의 CDRH3, SEQ ID NO: 148-164의 CDRH2 및 SEQ ID NO: 136-147의 CDRHl들을 포함하거나 및/또는 상기 단리된 항원 결합 단백질의 상기 제2아미노산 시퀀스는 SEQ ID NO: 225-246의 CDRL3, SEQ ID NO: 211-224의 CDRL2 및 SEQ ID NO: 191-210의 CDRLl들을 포함한다.
또 다른 구체예에 있어서, 상기 항원 결합 단백질은 표 4a에 나타낸 바와 같은 중쇄 시퀀스들 Hl, H2, H3, H4, H5, H6, H7, H8, H9, H1O, H11, H12, H13, H14, H15, H16, H17, H18, H19, H20, H21, H22, H23, H24, H25, H26, H27, H28, H29, H30, H31 또는 H32 중의 적어도 2개의 CDRH 시퀀스들을 포함한다. 다시 또 다른 구체예에 있어서, 상기 항원 결합 단백질은 표 4b에 나타낸 바와 같은 경쇄 시퀀스들 Ll, L2, L3, L4, L5, L6, L7, L8, L9, L1O, L11, L12, L13, L14, L15, L16, L17, L18, L19, L20, L21, L22, L23, L24, L25, L26, L27, L28, L29, L30, L31, L32, L33 또는 L34 중의 적어도 2개의 CDRL 시퀀스들을 포함한다. 다시 또 다른 구체예에 있어서, 상기 항원 결합 단백질은 표 4a에 나타낸 바와 같은 중쇄 시퀀스들 Hl, H2, H3, H4, H5, H6, H7, H8, H9, H1O, H11, H12, H13, H14, H15, H16, H17, H18, H19, H20, H21, H22, H23, H24, H25, H26, H27, H28, H29, H30, H31 또는 H32 중의 적어도 2개의 CDRH 시퀀스들 및 표 4b에 나타낸 바와 같은 경쇄 시퀀스들 Ll, L2, L3, L4, L5, L6, L7, L8, L9, L1O, L11, L12, L13, L14, L15, L16, L17, L18, L19, L20, L21, L22, L23, L24, L25, L26, L27, L28, L29, L30, L31, L32, L33 또는 L34 중의 적어도 2개의 CDRL 시퀀스들을 포함한다.
다시 다른 구체예에 있어서, 상기 항원 결합 단백질은 표 4a에 나타낸 바와 같은 Hl, H2, H3, H4, H5, H6, H7, H8, H9, H1O, H11, H12, H13, H14, H15, H16, H17, H18, H19, H20, H21, H22, H23, H24, H25, H26, H27, H28, H29, H30, H31 또는 H32 중의 상기 CDRHl, CDRH2 및 CDRH3 시퀀스들을 포함한다.
또 다른 구체예에 있어서, 상기 항원 결합 단백질은 표 4b에 나타낸 바와 같은 경쇄 시퀀스들 Ll, L2, L3, L4, L5, L6, L7, L8, L9, L1O, L11, L12, L13, L14, L15, L16, L17, L18, L19, L20, L21, L22, L23, L24, L25, L26, L27, L28, L29, L30, L31, L32, L33 또는 L34 중의 상기 CDRLl, CDRL2 및 CDRL3 시퀀스들을 포함한다.
또 다른 구체예에 있어서, 상기 항원 결합 단백질은 표 4a 및 표 4b들에 나타낸 바와 같은 Hl 및 Ll, 또는 Hl 및 L2, 또는 H2 및 L31, 또는 H2 및 L33, 또는 H3 및 L3, 또는 H4 및 L4, 또는 H5 및 L5, 또는 H6 및 L6, 또는 H7 및 L7, 또는 H8 및 L8, 또는 H8 및 L9, 또는 H9 및 L32, 또는 H9 및 L34, 또는 HlO 및 LlO, 또는 H11 및 L11, 또는 H12 및 L12, 또는 H13 및 L12, 또는 H14 및 L13, 또는 H15 및 L14, 또는 H16 및 L15, 또는 H17 및 L16, 또는 H18 및 L17, 또는 H19 및 L18, 또는 H20 및 L20, 또는 H21 및 L19, 또는 H22 및 L21, 또는 H24 및 L22, 또는 H25 및 L23, 또는 H26 및 L24, 또는 H27 및 L25, 또는 H28 및 L26, 또는 H29 및 L27, 또는 H30 및 L28, 또는 H31 및 L29, 또는 H32 및 L30의 6개의 CDRs들 모두를 포함한다.
이하의 실시예들에서 기술되는 바와 같이 제조되고 동정된 특정의 항체들에 대한 시퀀스 정보를 표 4c에 요약하였다. 대조의 편의를 위하여, 일부 경우들에 있어서, 대조번호의 축약된 형태가 본 명세서에서 사용되며, 여기에서 대조번호의 마지막 숫자를 탈락시켰다. 따라서, 예를 들면, 1.109.1은 때로 단순히 1.109로 언급하고; 1.109.1 SM은 1.109 SM으로 언급하고; 1.2.1은 1.2로 언급하고; 1.2.1 SM은 1.2 SM으로 언급하고; 2.360.1은 2.360으로 언급하고, 2.360.1 SM은 2.360 SM 등과 같이 언급한다.
[표 4a-1]
[표 4a-2]
[표 4b-1]
[표 4b-2]
[표 4c-1]
[표 4c-2]
하나의 관점에 있어서, 본 명세서에서 제공되는 상기 단리된 항원-결합 단백질들은 모노클로날 항체, 폴리클로날 항체, 재조합 항체, 인간 항체, 인간화된 항체, 키메라 항체, 다중특이성 항체 또는 이들의 항체 단편이 될 수 있다.
다른 구체예에 있어서, 본 명세서에서 제공되는 상기 단리된 항원-결합 단백질들의 상기 항체 단편은 Fab 단편, Fab' 단편, F(ab')2 단편, Fv 단편, 다이어바디 또는 단일쇄 항체 분자가 될 수 있다.
또 다른 구체예에 있어서, 본 명세서에서 제공되는 상기 단리된 항원 결합 단백질은 인간 항체이며, IgGl-형, IgG2-형, IgG3-형 또는 IgG4-형이 될 수 있다.
다른 구체예에 있어서, 상기 항원 결합 단백질은 단지 표 4a 내지 표 4c들에서 규정된 바와 같은 경쇄 및 중쇄 폴리펩티드로 이루어진다. 일부 구체예들에 있어서, 상기 항원 결합 단백질은 단지 표 4a 내지 표 4c들에서 규정된 바와 같은 가변 경쇄 도메인 및 가변 중쇄 도메인으로 이루어진다. 이러한 항원 결합 단백질들은 하나 또는 그 이상의 PEG 분자들로 피이지화(pegylated)될 수 있다.
또 다른 관점에 있어서, 본 명세서에서 제공되는 상기 단리된 항원-결합 단백질은 표지화기(labeling group)에 결합될 수 있으며, 또한 인간 c-fms의 세포외 부분에의 결합에 대하여 본 명세서에서 제공되는 상기 단리된 항원-결합 단백질들 중의 하나의 항원 결합 단백질과 경쟁할 수 있다. 하나의 구체예에 있어서, 본 명세서에서 제공되는 상기 단리된 항원 결합 단백질은 환자에 투여되는 경우에 단핵구 주화성을 감소시키고, 종양들 내로의 단핵구 이동을 저해하고, 종양 내에서의 종양 관련 마크로파지의 축적 및 기능을 저해할 수 있다.
당해 기술분야에서 통상의 지식을 가진 자에게는 이해될 수 있는 바와 같이, 상기 묘사된 시퀀스들로부터의 하나 이상의 CDR을 갖는 임의의 항원 결합 단백질에 대하여, 상기 묘사된 시퀀스들로부터 독립적으로 선택되는 CDRs들의 임의의 조합이 유용하다. 따라서, 1, 2, 3, 4, 5 또는 6개의 독립적으로 선택되는 CDRs들을 갖는 항원 결합 단백질들이 생성될 수 있다. 그러나, 당해 기술분야에서 통상의 지식을 가진 자에게는 이해될 수 있는 바와 같이, 특정의 구체예들은 대체로 비-반복적인 CDRs들의 조합을 활용하며, 예를 들면, 항원 결합 단백질들은 대체로 2개의 CDRH2 영역들 등으로 만들어지지는 않는다.
제공되는 상기 항원 결합 단백질들의 일부를 이하에서 보다 상세하게 설명한다.
항원 결합 단백질들 및 결합 에피토프들 및 바인딩 도메인들
항원 결합 단백질이 c-fms 등과 같은 특정화된 잔기들 또는 예를 들면 c-fms의 상기 세포외 도메인 내의 에피토프에 결합된다고 언급되는 경우, 이것이 의미하는 것은 상기 항원 결합 단백질이 c-fms의 특정의 부분에 특이적으로 결합한다는 것이다. 일부 구체예들에 있어서, 상기 항원 결합 단백질은 특정의 잔기들(예를 들면, c-fms의 특정의 세그먼트)로 이루어지는 폴리펩티드에 특이적으로 결합한다. 이러한 항원 결합 단백질은 전형적으로 c-fms 또는 c-fms의 상기 세포외 도메인의 모든 잔기와 접촉하지는 않는다. c-fms 또는 c-fms의 상기 세포외 도메인 내에서 모든 단일 아미노산 치환 또는 탈락되지는 않으며, 필수적으로 명백하게 바인딩 친화도에 영향을 준다.
항원 결합 단백질의 에피토프 특이성 및 상기 바인딩 도메인(들)은 다양한 방법들에 의해 결정될 수 있다. 예를 들면, 일부 방법들은 항원의 끝이 절단된 부분들을 사용할 수 있다. 다른 방법들은 하나 또는 그 이상의 특정의 잔기들에서 변이된 항원을 활용한다.
항원의 끝이 절단된 부분들을 사용하는 방법들에 대하여는, 하나의 예시적인 접근법에 있어서, 중첩 펩티드(overlapping peptides)들의 수집이 사용될 수 있다. 상기 중첩 펩티드들은 상기 항원의 상기 시퀀스에 걸치며 또한 적은 수의 아미노산들의 증분(increments)(예를 들면, 3개의 아미노산들)에서의 차이가 있는 대략 15개의 아미노산들로 이루어진다. 상기 펩티드들은 미세적정 접시(microtiter dish)의 벽들 내에 막 상의 서로 다른 위치들에서 부동태화된다. 부동태화는 상기 펩티드들의 하나의 말단의 비오티닐화(biotinylating)에 의해 발휘될 수 있다. 선택적으로, 상기 동일한 펩티드의 다른 샘프들이 아미노-말단 및 카르복시-말단에서 비오티닐화되고 그리고 비교의 목적을 위하여 별도의 벽들 내에서 부동태화될 수 있다. 이는 말단-특이적 항원 결합 단백질들의 동정에 유용하다. 선택적으로, 별도의 펩티드들이 대상체의 특정의 아미노산에서의 종결(terminating)에 포함될 수 있다. 이러한 접근법은 c-fms(또는 c-fms의 상기 세포외 도메인)의 내부 단편들에 대한 말단-특이적 항원 결합 단백질들의 동정에 유용하다. 상기 여러 펩티드들 각각의 특이적인 결합에 대하여 항원 결합 단백질 또는 면역학적으로 기능적인 단편이 스크리닝된다. 상기 에피토프는 상기 항원 결합 단백질이 특이적인 결합을 나타내는 모든 펩티드들에 대하여 공통인 아미노산들의 단편과 함께 발생하는 것으로 정의된다. 에피토프를 정의하기 위한 특이적인 접근법에 관한 상세를 실시예 12에서 규정하였다.
실시예 12에서 입증되는 바와 같이, 하나의 구체예에 있어서 본 명세서에서 제공되는 상기 항원 결합 단백질들은 Ig-형 도메인 1 및 Ig-형 도메인 2들을 함께 포함하는 폴리펩티드를 결합할 수 있으나; 그러나, 이들은 일차적으로는 상기 Ig-형 도메인 1 또는 일차적으로는 상기 Ig-형 도메인 2 단독을 포함하는 폴리펩티드를 결합하지는 않는다. 따라서 이러한 항원 결합 단백질들의 상기 결합 에피토프들은 상기 Ig-형 1 및 Ig-형 2 도메인들로 함께 하여 3차원적으로 구성된다. 도 8에서 강조된 바와 같이, 이들 두 도메인들은 c-fms 세포외 도메인의 아미노산 20-223들을 포함하며, 이는 하기의 아미노산 시퀀스를 갖는다:
IPVIEPSVPELVVKPGATVTLRCVGNGSVEWDGPPSPHWTLYSDGSSSILSTNNATFQNTGTYRCTEPGDPLGGSAAIHLYVKDPARPWNVLAQEVVVFEDQDALLPCLLTDPVLEAGVSLVRVRGRPLMRHTNYSFSPWHGFTIHRAKFIQSQDYQCSALMGGRKVMSISIRLKVQKVIPG PP ALTLVP ALVRIRGEAAQIV. (SEQ ID NO: 326)
상기 Ig-형 1 도메인을 나타내는 실시예 12에서 사용된 상기 아미노산 시퀀스는 도 8(즉, SEQ ID NO: 1의 아미노산들, 이른바 IPVIEPSVPELVVKPGATVTLRCVGNGSVEWDGPPSPHWTLYSDGSSSILSTNNATFQNTGTYRCTEPGDPLGGSAAIHLYVKDPARPWNVLAQEVVVFEDQDALLP)에 묘사된 상기 시퀀스의 아미노산 20-126)에 대응한다. 상기 Ig-형 2 도메인 단독을 나타내는 데 사용된 상기 아미노산 시퀀스는 도 8(즉, SEQ ID NO: 1의 아미노산들, 이른바 TEPGDPLGGSAAIHLYVKDPARPWNVLAQEVVVFEDQDALLPCLLTDPVLEAGVSLVRVRGRPLMRHTNYSFSPWHGFTIHRAKFIQSQDYQCSALMGGRKVMSISIRLKVQKVIPGPPALTLVPALVRIRGEAAQIV)에 묘사된 상기 시퀀스의 아미노산 85-223)에 대응한다.
따라서, 하나의 구체예에서의 항원 결합 단백질은 cfms 단백질(예를 들면, 성숙한 전장의 단백질) 내의 영역에 결합하거나 또는 특이적으로 결합할 수 있으며, 여기에서 상기 영역은 SEQ DO NO: 326에서 특정된 아미노산 시퀀스를 갖는다. 일부 구체예들에 있어서, 상기 항원 결합 단백질은 SEQ ED NO: 326에서 규정된 바와 같은 아미노산 잔기들로 필수적으로 이루어지거나 또는 이루어지는 폴리펩티드에 결합하거나 또는 특이적으로 결합한다.
다른 구체예에 있어서, 상기 항원 결합 단백질은 SEQ ID NO: 326으로 이루어지는 폴리펩티드에는 결합하거나 또는 특이적으로 결합할 수 있으나, 도 8에 묘사된 상기 시퀀스의 아미노산 20-126(즉, SEQ ID NO: 1의 아미노산 20-126)으로 이루어지는 폴리펩티드에는 그러하지 아니하다. 다른 관점에 있어서, 상기 항원 결합 단백질은 SEQ ID NO: 326으로 이루어지는 폴리펩티드에는 결합하거나 또는 특이적으로 결합할 수 있으나, 도 8에 묘사된 상기 시퀀스의 아미노산 85-223(즉, SEQ ID NO: 1의 아미노산 85-223)으로 이루어지는 폴리펩티드에는 그러하지 아니하다. 또 다른 구체예에 있어서, 상기 항원 결합 단백질은 SEQ ID NO: 326으로 이루어지는 폴리펩티드에는 결합하거나 또는 특이적으로 결합할 수 있으나, 도 8에 묘사된 상기 시퀀스의 아미노산 20-126(즉, SEQ ID NO: 1의 아미노산 20-126)으로 이루어지는 폴리펩티드 또는 도 8에 묘사된 상기 시퀀스의 아미노산 85-223(즉, SEQ ID NO: 1의 아미노산 85-223)으로 이루어지는 폴리펩티드에는 그러하지 아니하다.
다른 접근법에 있어서, 항체와 접촉하거나 또는 항체에 의해 매립되는 잔기들을 포함하는 상기 도메인(들)/영역(들)은 항원(예를 들면, 야생형(wild type) 항원) 내의 특정의 잔기들을 돌연변이시키고 그리고 상기 항원 결합 단백질이 돌연변이된 단백질에 결합할 수 있는 지의 여부를 결정하는 것에 의하여 동정될 수 있다. 다수의 개개 돌연변이들을 만드는 것에 의하여, 결합에서 직접적인 역할을 하거나 또는 돌연변이가 상기 항원 결합 단백질과 항원 사이의 결합에 영향을 줄 수 있도록 상기 항체에 충분히 가깝게 접근하는 잔기들이 동정될 수 있다. 이들 아미노산들의 지식으로부터, 상기 항원 결합 단백질과 접촉하거나 또는 상기 항체에 의해 덮여지는 잔기들을 포함하는 상기 항원의 상기 도메인(들) 또는 영역(들)이 밝혀질 수 있다. 이러한 도메인은 전형적으로 항원 결합 단백질의 결합 에피토프를 포함한다. 이러한 일반적인 접근법의 하나의 특정의 구체예는 아르기닌/글루타민산 주사 프로토콜(arginine/glutamic acid scanning protocol)을 활용한다(예를 들면, 나네비츠, 티.와 그의 동료들(Nanevicz, T., et al)의 1995, J. Biol. Chem., 270:37, 21619-21625 및 주프니크, 에이와 그의 동료들(Zupnick, A., et al)의 2006, J. Biol. Chem., 281:29, 20464-20473들을 참조하시오). 대체로, 아르기닌 및 글루타민산들은 하전되고 그리고 부피가 크며(bulky) 따라서 돌연변이가 도입된 상기 항원의 상기 영역 내에서의 항원 결합 단백질과 항원 사이의 결합을 방해하는 잠재성을 갖기 때문에 이들 아미노산들은 야생형 폴리펩티드 내에서의 아미노산으로 대체(전형적으로는 개별적으로) 된다. 야생형 항원 내에 존재하는 아르기닌 및 리신들이 글루타민산으로 대체된다. 이러한 다양한 개별적인 돌연변이들을 수득하고 그리고 수집된 결합 결과들을 분석하여 어떤 잔기들이 결합에 영향을 주는 지를 결정한다.
실시예 14는 본 명세서에서 제공되는 c-fms 결합 단백질들에 대한 인간 c-fms의 아르기닌/글루타민산 주사를 기술하고 있다. 단일의 돌연변이를 갖는 각 돌연변이 항원으로 일련의 95개의 돌연변이 인간 c-fms 항원들이 생성되었다. 각 돌연변이 c-fms 항원의 본 명세서에서 제공되는 선택된 c-fms 항원 결합 단백질들과의 결합을 측정하고 그리고 이들 선택된 결합 단백질들이 야생형 c-fms 항원(SEQ ID NO: 1)에 결합하는 능력과 비교하였다. 항원 결합 단백질과 본 명세서에서 사용된 바와 같은 돌연변이 c-fms 항원 사이의 결합에서의 감소는 결합 친화도(예를 들면, 상기 실시예들에서 기술된 바와 같은 바이어코어 시험(Biacore testing) 등과 같은 공지의 방법들에 의해 측정된 바와 같은) 및/또는 상기 항원 결합 단백질의 총 결합용량(total binding capacity)(예를 들면, 항원 결합 단백질 농도 대 항원 농도의 곡선(plot)에서의 Bmax에서의 감소에 의해 증거되는 바와 같이)에서의 감소가 있다는 것을 의미한다. 결합에서의 명백한 감소는 상기 돌연변이된 잔기가 상기 항원 결합 단백질에의 결합에 직접적으로 참여하거나 또는 상기 결합 단백질이 항원에 부착되는 경우에 상기 결합 단백질에 긴밀하게 근접한다는 것을 나타내고 있다.
일부 구체예들에 있어서, 결합에서의 명백한 감소는 항원 결합 단백질과 돌연변이 c-fms 항원 사이의 결합 친화도 및/또는 용량이 상기 결합 단백질과 야생형 c-fms 항원(예를 들면, SEQ ID NO: 1에 나타낸 상기 세포외 도메인) 사이의 결합에 대하여 40% 이상, 50% 이상, 55% 이상, 60% 이상, 65% 이상, 70% 이상, 75% 이상, 80% 이상, 85% 이상, 90% 이상 또는 95% 이상으로 감소된다는 것을 의미한다. 특정의 구체예들에 있어서, 결합은 검출가능한 한계들 이하로 감소된다. 일부 구체예들에 있어서, 항원 결합 단백질의 돌연변이 c-fms 항원에의 결합이 상기 항원 결합 단백질과 야생형 c-fms 항원(예를 들면, SEQ ID NO: 1에 나타낸 상기 세포외 도메인) 간에서 관측된 결합의 50% 이하(예를 들면, 45%, 40%, 35%, 30%, 25%, 20%, 15% 또는 10% 이하) 인 경우에서 결합에서의 명백한 감소가 입증되었다. 이러한 결합 측정들은 당해 기술분야에서 공지된 다양한 결합 분석법들을 사용하여 이루어질수 있다. 하나의 이러한 분석법의 특정의 예가 실시예 14에서 기술된다.
일부 구체예들에 있어서, 야생형 c-fms 항원(예를 들면, SEQ ED NO: 1) 내의 잔기가 아르기닌 또는 글루타민산으로 치환된 돌연변이 c-fms 항원에 대한 명백하게 낮은 결합을 나타내는 항원 결합 단백질들이 제공된다. 하나의 이러한 구체예에 있어서, 항원 결합 단백질의 결합은 야생형 c-fms(예를 들면, SEQ ED NO: 1)에 비하여 하기의 돌연변이들 즉 E29R, Q121R, T152R 및 K185E들 중의 임의의 하나 또는 그 이상을 갖는 돌연변이 c-fms 항원에 대하여 명백하게 감소된다. 본 명세서에서 사용되는 속기표기(shorthand notation)에 있어서, 그 형식은 SEQ ID NO: 1에서 표시된 바와 같은 상기 잔기들의 번호들과 함께 야생형 잔기:폴리펩티드 내에서의 위치:돌연변이 잔기이다. 일부 구체예들에 있어서, 항원 결합 단백질의 결합은 야생형 c-fms(예를 들면, SEQ ED NO: 1)에 비하여 하기의 돌연변이들 즉, E29R, Q121R, S172R, G274R 및 Y276R들 중의 임의의 하나 또는 그 이상(예를 들면, 1, 2, 3, 4 또는 5)을 갖는 돌연변이 c-fms 항원에 대하여 명백하게 감소된다. 다른 구체예에 있어서, 항원 결합 단백질은 야생형 c-fms(예를 들면, SEQ ED NO: 1)에 비하여 하기의 돌연변이들 즉, R106E, H151R, T152R, Y154R, S155R, W159R, Q171R, S172R, Q173R, G183R, R184E, K185E, E218R, A220R, S228R, H239R, N240R, K259E, G274R, N275R, Y276R, S277R 및 N282R들 중의 임의의 하나 또는 그 이상(예를 들면, 1, 2, 3, 4, 5 등 23까지)을 갖는 돌연변이 c-fms 항원에 대하여 명백하게 낮은 결합을 나타낸다. 심지어 또 다른 구체예들에 있어서, 항원 결합 단백질의 결합은 야생형 c-fms(예를 들면, SEQ ED NO: 1)에 비하여 하기의 돌연변이들 즉, K102E, R144E, R146E, Dl 74R 및 A226R들 중의 임의의 하나 또는 그 이상(예를 들어, 1, 2, 3, 4 또는 5)을 갖는 돌연변이 c-fms 항원에 대하여 명백하게 감소된다. 또 다른 구체예들에 있어서, 항원 결합 단백질은 야생형 c-fms(예를 들면, SEQ ED NO: 1)에 비하여 하기의 돌연변이들 즉, W50R, A74R, YlOOR, D122R, T130R, G161R, Y175R 및 A179R들 중의 임의의 하나 또는 그 이상(예를 들어, 1, 2, 3, 4, 5, 6, 7 또는 8)을 갖는 돌연변이 c-fms 항원에 대하여 명백하게 감소된 결합을 나타낸다.
비록 SEQ ID NO: 1에 나타낸 야생형 세포외 도메인 시퀀스에 대하여 앞서 기재된 상기 돌연변이 형태들을 검토하였으나, c-fms의 대립형질적 변종(allelic variant)들에 있어서 표시된 위치에서의 상기 아미노산은 다를 수 있다는 것은 이해될 수 있는 것이다. c-fms의 이러한 대립형질적 형태들에 대한 명백하게 낮은 결합을 나타내는 항원 결합 단백질들이 또한 고려된다. 따라서, 하나의 구체예에 있어서, 항원 결합 단백질은 야생형 c-fms(예를 들면, SEQ ID NO: 1)에 비하여 대립형질적 c-fms 항원에 대하여 명백하게 감소된 결합을 가지며, 여기에서 대립형질적 항원의 하기의 잔기들의 하나 또는 그 이상(예를 들면, 1, 2, 3 또는 4)이 하기 표시된 바와 같이 아르기닌 또는 글루타민산으로 대체된다: 29R, 121R, 152R 및 185E(SEQ ID NO: 1에서 표시된 바와 같은 상기 잔기들의 번호와 함께의 폴리펩티드의 위치: 돌연변이 잔기). 일부 구체예들에 있어서, 항원 결합 단백질은 야생형 c-fms(예를 들면, SEQ ID NO: 1)에 결합하는 능력에 비하여 하기의 잔기들의 하나 또는 그 이상(예를 들면, 1, 2, 3, 4 또는 5)이 하기 표시된 바와 같이 아르기닌 또는 글루타민산으로 대체되는 대립형질적 c-fms 항원에 대하여 명백하게 감소된 결합을 나타낸다: 29R, 121R, 172R, 274R 및 276R. 또 다른 구체예에 있어서, 항원 결합 단백질은 야생형 c-fms(예를 들면, SEQ ID NO: 1)에 결합하는 능력에 비하여 하기의 잔기들의 하나 또는 그 이상(예를 들면, 1, 2, 3, 4, 5 등 23까지)이 하기 표시된 바와 같이 아르기닌 또는 글루타민산으로 대체되는 대립형질적 c-fms 항원에 대하여 명백하게 감소된 결합을 나타낸다: 106E, 151R, 152R, 154R, 155R, 159R, 171R, 172R, 173R, 183R, 184E, 185E, 218R, 220R, 228R, 239R, 240R, 259E, 274R, 275R, 276R, 277R 및 282R. 또 다른 구체예들에 있어서, 항원 결합 단백질은 야생형 c-fms(예를 들면, SEQ ID NO: 1)에 결합하는 능력에 비하여 하기의 잔기들의 하나 또는 그 이상(예를 들면, 1, 2, 3, 4 또는 5)이 하기 표시된 바와 같이 아르기닌 또는 글루타민산으로 대체되는 대립형질적 c-fms 항원에 대하여 명백하게 감소된 결합을 나타낸다: 102E, 144E, 146E, 174R 및 226R. 또 다른 구체예들에 있어서, 항원 결합 단백질은 야생형 c-fms(예를 들면, SEQ ID NO: 1)에 결합하는 능력에 비하여 하기의 잔기들의 하나 또는 그 이상(예를 들면, 1, 2, 3, 4, 5, 6, 7 또는 8)이 하기 표시된 바와 같이 아르기닌 또는 글루타민산으로 대체되는 대립형질적 c-fms 항원에 대하여 명백하게 감소된 결합을 나타낸다: 5OR, 74R, 10OR, 122R, 13OR, 161R, 175R 및 179R.
일부 구체예들에 있어서, 항원 결합 단백질의 결합은 상기 야생형 c-fms 항원 내의 선택된 위치에서의 상기 잔기가 임의의 다른 잔기로 돌연변이되는 돌연변이 c-fms 항원에 대하여 명백하게 감소된다. 예를 들면, 하나의 구체예에 있어서, 항원 결합 단백질은 야생형 c-fms(예를 들면, SEQ ID NO: 1)에 결합하는 능력에 비하여 위치 29, 121, 152 및 185들(여기에서 상기 위치들은 SEQ ID NO: 1에서 표시된 것과 마찬가지이다) 중의 하나 또는 그 이상(예를 들면, 1, 2, 3 또는 4)에서 단일의 아미노산 치환을 포함하는 돌연변이 c-fms 항원에 대하여 명백하게 감소된 결합을 나타낸다. 일부 구체예들에 있어서, 항원 결합 단백질은 야생형 c-fms(예를 들면, SEQ ID NO: 1)에 결합하는 능력에 비하여 SEQ ID NO: 1의 위치 29, 121, 172, 274 및 276들 중의 하나 또는 그 이상(예를 들면, 1, 2, 3, 4 또는 5)에서 단일의 아미노산 치환을 포함하는 돌연변이 c-fms 항원에 대하여 명백하게 감소된 결합을 갖는다. 다른 구체예에 있어서, 항원 결합 단백질의 결합은 SEQ ID NO: 1의 야생형 c-fms에의 결합에 비하여 SEQ ID NO: 1의 위치 106, 151, 152, 154, 155, 159, 171, 172, 173, 183, 184, 185, 218, 220, 228, 239, 240, 259, 274, 275, 276, 277 및 282들 중의 하나 또는 그 이상(예를 들면, 1, 2, 3, 4, 5 등 23까지)에서 단일의 아미노산 치환을 포함하는 돌연변이 c-fms 항원에 대하여 명백하게 감소된다. 또 다른 구체예들에 있어서, 항원 결합 단백질은 야생형 c-fms(예를 들면, SEQ ID NO: 1)에 결합하는 능력에 비하여 SEQ ID NO: 1의 위치 102, 144, 146, 174 및 226들 중의 하나 또는 그 이상(예를 들면, 1, 2, 3, 4 또는 5)에서 단일의 아미노산 치환을 포함하는 돌연변이 c-fms 항원에 대하여 명백하게 낮은 결합을 갖는다. 또 다른 구체예들에 있어서, 항원 결합 단백질은 야생형 c-fms(예를 들면, SEQ ID NO: 1)에 결합하는 능력에 비하여 위치 50, 74, 100, 122, 130, 161, 175 및 179들 중의 하나 또는 그 이상(예를 들면, 1, 2, 3, 4, 5, 6, 7 또는 8)에서 단일의 아미노산 치환을 포함하는 돌연변이 c-fms 항원에 대한 명백하게 낮은 결합을 갖는다.
앞서 언급한 바와 같이, 결합에 직접적으로 참여하거나 또는 항원 결합 단백질에 의하여 덮여지는 잔기들은 주사 결과들로부터 동정될 수 있다. 따라서 이들 잔기들은 항원 결합 단백질들이 결합하는 결합 영역(들)을 포함하는 SEQ ID NO: 1의 도메인들 또는 영역들의 표시를 제공할 수 있다. 실시예 14에서 요약된 결과들로부터 알 수 있는 바와 같이, 하나의 구체예에 있어서, 항원 결합 단백질은 SEQ ID NO: 1의 아미노산 29-185들을 포함하는 도메인에 결합한다. 다른 구체예에 있어서, 상기 항원 결합 단백질은 SEQ ID NO: 1의 아미노산 29-276들을 포함하는 영역에 결합한다. 다른 구체예들에 있어서, 상기 항원 결합 단백질은 SEQ ID NO: 1의 아미노산 106-282들을 포함하는 영역에 결합한다. 또 다른 구체예들에 있어서, 상기 항원 결합 단백질은 SEQ ID NO: 1의 아미노산 102-226들을 포함하는 영역에 결합한다. 또 다른 구체예들에 있어서, 상기 항원 결합 단백질은 SEQ ID NO: 1의 아미노산 50-179들을 포함하는 영역에 결합한다. 일부 구체예들에 있어서, 상기 항원 결합 단백질들은 SEQ ID NO: 1의 전장 시퀀스의 단편 내의 앞서의 영역들에 결합한다. 다른 구체예들에 있어서, 항원 결합 단백질들은 이들 영역들로 이루어지는 폴리펩티드들에 결합한다. 특정의 구체예들에 있어서, 항원 결합 단백질은 SEQ ID NO: 1의 아미노산 90-282들을 포함하는 영역에 결합한다. 또 다른 구체예에 있어서, 항원 결합 단백질은 SEQ ID NO: 1의 아미노산 90-185들 및 아미노산 217-282들의 영역들 중의 하나 또는 둘 다에 결합한다. 또 다른 구체예에 있어서, 항원 결합 단백질은 SEQ ID NO: 1의 아미노산 121-185들 및 아미노산 217-277들의 영역들 중의 하나 또는 둘 다에 결합한다.
경쟁하는 항원 결합 단백질들
다른 관점에 있어서, c-fms에의 특정의 결합에 대하여 앞서 기술된 상기 에피토프에 결합하는 예시화된 항체들 또는 기능적인 단편들 중의 하나와 경쟁하는 항원 결합 단백질들이 제공된다. 이러한 항원 결합 단백질들은 또한 본 명세서에서 예시화된 항원 결합 단백질들 또는 중첩 에피토프들과 동일한 에피토프에 결합할 수 있다. 상기 예시화된 항원 결합 단백질들과 동일한 에피토프와 경쟁하거나 또는 그에 결합하는 항원 결합 단백질들 및 단편들은 동일한 기능적인 특성들을 나타낼 것으로 기대된다. 상기 예시화된 항원 결합 단백질들 및 단편들은 표 1, 표 2, 표 3 및 표 4a 내지 표 4c들 내에 포함된 중쇄 및 경쇄들, 가변영역 도메인들 및 CDRs들을 포함하여 앞서 기술한 것들을 포함한다. 따라서, 특정의 구체예로서, 제공되는 상기 항원 결합 단백질들은 다음의 것들을 갖는 항체와 경쟁하는 것들을 포함한다:
(a) 표 4c에 기재된 항체에 대해 기재된 상기 CDRs 6개 전부;
(b) 표 4c에 기재된 항체에 대해 기재된 VH 및 VL; 또는
(c) 표 4c에기재된 항체에 대해 특정화된 것으로서의 2개의 경쇄들 및 2개의 중쇄들.
모노클로날 항체
제공되는 상기 항원 결합 단백질들은 c-fms에 결합하는 모노클로날 항체들을 포함한다. 모노클로날 항체들은 당해 기술분야에서 공지된 임의의 기술을 사용하여, 예를 들어, 면역화 스케쥴(immunization schedule) 후에 이식유전자를 가진 동물(transgenic animal)로부터 수확된 비장 세포들의 불멸화(immortalizing)에 의하여 생산될 수 있다. 상기 비장 세포들은 당해 기술분야에서 공지된 임의의 기술을 사용하여, 예를 들어, 이들을 골수종 세포(myeloma cells)들과 융합시켜 하이브리도마를 생성하는 것에 의하여 불멸화될 수 있다. 하이브리도마-생성 융합 절차들에서 사용되기 위한 골수종 세포들은 바람직하게는 비-항체-생성이고, 높은 융합 효율을 가지고, 그리고 원하는 융합된 세포(하이브리도마)들의 성장을 뒷받침하는 특정의 선택 매질(media) 내에서의 이들의 성장을 불가능하게 하는 효소 결핍된 것들이다. 생쥐 융합들에서 사용하기에 적절한 세포주들의 예들에는 Sp-20, P3-X63/Ag8, P3-X63-Ag8.653, NSl/l.Ag 4 1, Sp210-Agl4, FO, NSO/U, MPC-11, MPC11-X45-GTG 1.7 및 S194/5XXO Bul들이 포함되고; 집쥐 융합들에서 사용되는 세포주들의 예들에는 R210.RCY3, Y3-Ag 1.2.3, IR983F 및 4B210들이 포함된다. 세포 융합들에 사용될 수 있는 다른 세포주들은 U-266, GM1500-GRG2, LICR-LON-HMy2 및 UC729-6들이다.
일부 경우들에 있어서, 동물(예를 들면, 인간 면역글로블린 시퀀스들을 갖는 이식유전자를 가진 동물)을 c-fms로 면역화시키고; 상기 면역화된 동물로부터 비장 세포들을 수확하고; 상기 수확된 비장 세포들을 골수종 세포주에 융합시키고, 그에 의하여 하이브리도마 세포들을 생성시키고; 상기 하이브리도마 세포들로부터 하이브리도마 세포주들을 구축하고; 그리고 c-fms 폴리펩티드에 결합하는 항체를 생성하는 하이브리도마 세포주를 동정하는 것에 의하여 하이브리도마 세포주가 생성된다. 이러한 하이브리도마 세포주들 및 이들에 의해 생산되는 항-c-fms 모노클로날 항체들이 본 출원의 관점들이다.
하이브리도마 세포주에 의해 분비되는 모노클로날 항체들은 당해 기술분야에서 공지된 임의의 기술을 사용하여 정제될 수 있다. 하이브리도마들 또는 mAbs들은 더 스크리닝되어 Wnt 유도 활성을 차단하는 능력 등과 같은 특정의 특성들을 갖는 mAbs를 동정할 수 있다. 이러한 스크리닝의 예들이 이하의 실시예들에서 제공된다.
키메라 항체 및 인간화된 항체들
앞서의 시퀀스들에 기초하는 키메라 항체 및 인간화된 항체들이 또한 제공된다. 치료적인 약품들로서 사용되기 위한 모노클로날 항체들은 사용에 앞서 여러 방법들로 변형될 수 있다. 하나의 구체예는 키메라 항체이며, 이는 공유적으로 결합되어 기능적인 면역글로블린 경쇄 및 중쇄들 또는 이들의 면역학적으로 기능적인 부분들을 생산하는 다른 항체들로부터의 단백질 단편들로 이루어지는 항체이다. 대체로, 상기 중쇄 및/또는 경쇄의 일부는 특정의 종들로부터 유래되거나 또는 특정의 항체 클래스 또는 서브클래스에 속하는 항체들 내의 대응하는 시퀀스와 동일하거나 또는 상응하는 것인 반면에, 상기 쇄(들)의 잔여부(remainder)(들)은 다른 종들로부터 유래되거나 또는 다른 항체 클래스 또는 서브클래스에 속하는 항체들 내의 대응하는 시퀀스와 동일하거나 또는 상응하는 것이다. 키메라 항체들과 관련되는 방법들에 대하여는, 예를 들면, 본 명세서에서 참조로 인용하고 있는 미합중국 특허 제4,816,567호; 및 모리슨과 그의 동료들(Morrison et al)의 1985, Proc. Natl. Acad. Sci. USA 81:6851-6855들을 참조하시오. CDR 분지화(CDR grafting ; 접붙이기)는, 예를 들면, 미합중국 특허 제6,180,370호, 동 제5,693,762호, 동 제5,693,761호, 동 제5,585,089호 및 동 제5,530,101호들에 기술되어 있다.
대체로, 키메라 항체를 제조하는 목표는 목적하는 환자 종(patient species)으로부터의 아미노산들의 수가 최대가 되는 키메라를 생성하는 것이다. 하나의 구체예는 상기 "CDR-분지화된" 항체이며, 여기에서 상기 항체는 특정의 종들로부터의 또는 특정의 항체 클래스 또는 서브클래스에 속하는 하나 또는 그 이상의 상보적 결합 영역들(CDRs)을 포함하는 반면에, 상기 항체 쇄(들)의 잔여부(들)은 다른 종들로부터 유래되거나 또는 다른 항체 클래스 또는 서브클래스에 속하는 항체들 내의 대응하는 시퀀스와 동일하거나 또는 상응하는 것이다. 인간들에서 사용하기 위해서는, 설치류 항체(rodent antibody)로부터의 가변영역 또는 선택된 CDRs들이 종종 인간 항체 내로 분지되어 상기 인간 항체의 자연적으로 발생하는 가변영역들 또는 CDRs들을 대체한다.
키메라 항체의 하나의 유용한 형태는 "인간화된" 항체이다. 대체로, 인간화된 항체는 초기에 비-인간의 동물에서 야기되는 모노클로날 항체로부터 생산된다. 이 모노클로날 항체 내의 특정의 아미노산 잔기들, 전형적으로는 상기 항체의 비-항원 인식 부분들이 대응하는 아이소타입의 인간 항체 내의 대응하는 잔기들에 상응하도록 변형된다. 예를 들면, 인간화는 다양한 방법들을 사용하여 인간 항체의 대응하는 영역들에 대한 설치류의 가변영역의 적어도 일부를 치환하는 것에 의하여 수행될 수 있다(예를 들면, 미합중국 특허 제5,585,089호, 동 제5,693,762호; 존스와 그의 동료들(Jones et al.)의 1986, Nature 321:522-525; 라이만과 그의 동료들(Riechmann et al)의 1988, Nature 332:323-27; 베르호에옌과 그의 동료들(Verhoeyen et al.)의 1988, Science 239:1534-1536들을 참조하시오).
하나의 관점에 있어서, 본 명세서에서 제공되는 상기 항체들의 경쇄 및 중쇄 가변영역들의 상기 CDRs들(표 3을 참조하시오)은 동일하거나 또는 서로 다른 계통발생적인 종들로부터의 항체들로부터의 골격영역(FRs)에 분지된다. 예를 들면, 중쇄 및 경쇄 가변영역 VH1, VH2, VH3, VH4, VH5, VH6, VH7, VH8, VH9, VH10, VH11, VH12, VH13, VH14, VH15, VH16, VH17, VH18, VH19, VH20, VH21, VH22, VH23, VH24, VH25, VH26, VH27, VH28, VH29, VH30, VH31 및 VH32 및/또는 VL1, VL2, VL3, VL4, VL5, VL6, VL7, VL8, VL9, VL10, VL11, VL12, VL13, VL14, VL15, VL16, VL17, VL18, VL19, VL20, VL21, VL22, VL23, VL24, VL25, VL26, VL27, VL28, VL29, VL30, VL31, VL32, VL33 및 VL34들의 CDRs들이 합치 인간 FRs에 분지될 수 있다. 합치 인간 FRs를 생성하기 위해서는, 여러 인간 중쇄 또는 경쇄 아미노산 시퀀스들로부터의 FRs들을 정렬시켜 합치 아미노산 시퀀스를 동정하도록 할 수 있다. 다른 구체예들에 있어서, 본 명세서에서 기술된 중쇄 또는 경쇄의 상기 FRs들은 다른 중쇄 또는 경쇄로부터의 FRs들로 대체된다. 하나의 관점에 있어서, 항-c-fms의 중쇄 및 경쇄의 FRs들 내에서의 희귀한 아미노산들은 대체되지 않는 반면에, 상기 FR 아미노산들의 나머지는 대체된다. "희귀한 아미노산"은 이 특정의 아미노산이 RF 내에서 통상적으로 발견되지 않는 특정의 아미노산 내의 위치 안에 존재하는 특정의 아미노산이다. 달리, 하나의 중쇄 또는 경쇄로부터의 상기 분지된 가변영역들은 본 명세서에서 기술되는 바와 같은 특정의 중쇄 또는 경쇄의 불변영역과는 다른 불변영역과 함께 사용될 수 있다. 다른 구체예들에 있어서, 상기 분지된 가변영역들은 단일쇄 Fv 항체의 일부이다.
특정의 구체예들에 있어서, 인간 이외의 종들로부터의 불변영역들이 인간 가변영역(들)과 함께 사용되어 잡종 항체들을 생산할 수 있다.
완전한 인간 항체들
완전한 인간 항체들이 또한 제공된다. 인간을 상기 항원("완전한 인간 항체들")에 노출시킴이 없이 주어진 항원에 대하여 특이적인 완전한 인간 항체들을 제조하기 위한 방법들이 획득가능하다. 완전한 인간 항체들의 생산을 실행하기 위하여 제공되는 하나의 특정한 수단은 생쥐 체액성 면역 시스템(mouse humoral immune system)의 "인간화"이다. 내생 Ig 유전자들이 비활성화된 생쥐 내로의 인간 면역글로블린(Ig) 로커스들(loci)을 도입하는 것이 임의의 원하는 항원으로 면역화될 수 있는 동물인 생쥐 내에서 완전한 인간 모노클로날 항체들(mAbs)을 생산하는 하나의 수단이다. 완전한 인간 항체들을 사용하는 것은 때때로 치료적 약품들로서 생쥐 또는 생쥐-유래의 mAbs들을 인간들에 투여하는 것에 의해 야기될 수 있는 면역성 반응들 또는 알러지 반응들을 최소화시킬 수 있다.
완전한 인간 항체들은 내생 면역글로블린 생산이 없는 인간 항체의 전목록(repertoire)을 생산할 수 있는 이식유전자를 가진 동물들(대개 생쥐)을 면역화시키는 것에 의해 생산될 수 있다. 이러한 목적을 위한 항원들은 전형적으로 6개 또는 그 이상의 근접하는 아미노산들을 가지며, 선택적으로 부착소 등과 같은 담체(carrier)에 콘쥬게이트 된다. 예를 들면, 자코보비츠와 그의 동료들(Jakobovits et al)의 1993, Proc. Natl. Acad. Sci. USA 90:2551-2555; 자코보비츠와 그의 동료들의 1993, Nature 362:255-258; 및 브루거만과 그의 동료들(Bruggermann et al)의 1993, Year in Immunol. 7:33들을 참조하시오. 이러한 방법의 하나의 구체예에 있어서, 생쥐 중쇄 및 경쇄 면역글로블린(heavy and light immunoglobulin chains)들을 인코딩하는 내생 생쥐 면역글로블린 로커스를 무능화(incapacitating)시키고, 그리고 상기 생쥐 게놈 내로 인간 중쇄 및 경쇄 단백질들을 인코딩하는 인간 게놈 DNA의 거대 단편들을 삽입시키는 것에 의해 이식유전자를 가진 동물들이 생산된다. 계속해서 인간 면역글로블린 로커스의 완전한 보체(complement) 보다 작은 부분적으로 변형된 동물들을 이종교배(cross-bred)시켜 원하는 면역 시스템 변형들 모두를 갖는 동물이 수득된다. 면역원(immunogen)이 투여되는 경우, 이들 이식유전자를 가진 동물들은 상기 면역원에 대해 면역특이적이나 상기 가변영역들을 포함하여 쥣과의 아미노산 시퀀스들 보다는 오히려 인간 아미노산 시퀀스들을 갖는 항체들을 생산한다. 이러한 방법들의 보다 상세한 설명들을 위하여는, 예를 들면, 국제특허공개공보 제WO96/33735호 및 동 제WO94/02602호들을 참조하시오. 인간 항체들을 생산하기 위한 이식유전자를 갖는 생쥐과 관련한 부가의 방법들은 미합중국 특허 제5,545,807호, 동 제6,713,610호, 동 제6,673,986호, 동 제6,162,963호, 동 제5,545,807호, 동 제6,300,129호, 동 제6,255,458호, 동 제5,877,397호, 동 제5,874,299호 및 동 제No. 5,545,806호; 국제특허공개공보 제WO91/10741호, 동 제WO90/04036호; 및 유럽특허 제546073B1호 및 동 제546073Al호들에 기술되어 있다.
본 명세서에서 "HuMab" 생쥐로 언급되는 앞서 기술한 이식유전자를 갖는 생쥐는 내생 [뮤] 및 [카파] 쇄 로커스들을 비활성화시키는 목표로 하는 돌연변이들과 함께 재배열되지 않은 인간 중쇄([뮤] 및 [감마]) 및 [카파] 경쇄 면역글로블린 시퀀스들을 인코딩하는 인간 면역글로블린 유전자 미니로커스(minilocus)를 포함한다(론버그와 그의 동료들(Lonberg et al)의 1994, Nature 368:856-859). 따라서, 상기 생쥐는 생쥐 IgM 또는 [카파]의 감소된 발현을 나타내고, 그리고 면역화에 응하여 상기 도입된 인간 중쇄 및 경쇄 이식유전자(transgenes)들은 클래스 스위칭(class switching) 및 체세포적 돌연변이(somatic mutation)를 수행하여 높은 친화도의 인간 IgG [카파] 모노클로날 항체들을 생산한다(론버그와 그의 동료들의 상기 문헌; 론버그와 허스짜르(Lonberg and Huszar)의 1995, Intern. Rev. Immunol. 13:65-93; 하딩과 론버그(Harding and Lonberg)의 1995, Ann. NY Acad. Sci. 764:536-546). HmMab 생쥐의 제조는 테일러와 그의 동료들(Taylor et al)의 1992, Nucleic Acids Research 20:6287-6295; 첸과 그의 동료들의 1993, International Immunology 5:647-656; 투아일론(Tuaillon)과 그의 동료들의 1994, J. Immunol. 152:2912-2920; 론버그와 그의 동료들의 1994, Nature 368:856-859; 론버그의 1994, Handbook of Exp. Pharmacology 113:49-101 ; 테일러와 그의 동료들의 1994, International Immunology 6:579-591 ; 론버그와 허스짜르의 1995, Intern. Rev. Immunol. 13:65-93; 하딩과 론버그의 1995, Ann. NY Acad. Sci. 764:536-546; 피시와일드(Fishwild)와 그의 동료들의 1996, Nature Biotechnology 14:845-851;들에 상세하게 기술되어 있으며, 앞서의 참조문헌들은 모든 목적들을 위한 그들의 전체로 본 명세서에 참조로 인용된다. 또한, 다음의 참조문헌들이 모든 목적들을 위한 그들의 전체로 본 명세서에 참조로 인용되는 미합중국 특허 제5,545,807호; 국제특허공개공보 제WO 93/1227호; 동 제WO 92/22646호; 및 동 제WO 92/03918호들과 마찬가지로 미합중국 특허 제5,545,806호; 동 제5,569,825호; 동 제5,625,126호; 동 제5,633,425호; 동 제5,789,650호; 동 제5,877,397호; 동 제5,661,016호; 동 제5,814,318호; 동 제5,874,299호; 및 동 제5,770,429호들을 참조하시오. 이들 이식유전자를 갖는 생쥐에서 인간 항체들을 생산하는 데 활용되는 기술들은 또한 국제특허공개공보 제WO 98/24893호 및 멘데즈(Mendez)와 그의 동료들의 1997, Nature Genetics 15:146-156에 기술되며, 이들은 본 명세서에서 참조로 인용된다. 예를 들면, 상기 HCo7 및 HCo 12 이식유전자를 가진 생쥐 스트레인들은 항-c-fms 항체들을 생산하는 데 사용될 수 있다. 이식유전자를 가진 생쥐를 사용하는 인간 항체들의 생산과 관련한 보다 상세한 설명들을 이하에 예시적으로 제공한다.
하이브리도마 기술을 사용하여, 원하는 특이성을 갖는 항원-특이적인 인간 mAbs가 앞서 기술한 바와 같은 이식유전자를 가진 생쥐로부터 생산되고 그리고 선택될 수 있다. 이러한 항체들은 적절한 벡터 및 숙주세포들을 사용하여 복제되고 그리고 발현될 수 있거나 또는 상기 항체들은 배양된 하이브리도마 세포들로부터 수확될 수 있다.
완전한 인간 항체들은 또한 파지-디스플레이 라이브러리(phage-display libraries)들로부터 유도될 수 있다(후겐붐(Hoogenboom)과 그의 동료들의 1991, J. Mol. Biol. 227:381; 및 마크스(Marks)와 그의 동료들의 1991, J. Mol. Biol. 222:581)들에 기술된 바와 같음). 파지 디스플레이 기술은 심질의 박테리오파지(filamentous bacteriophage)의 표면에 대한 항체 전목록들의 디스플레이 및 후속의 선택되는 항원에의 그들의 결합에 의한 파지의 선택을 통한 면역 선택을 모방한다. 이러한 기술의 하나는 국제특허공개공보 제99/10494호(본 명세서에서 참조로 인용됨)에 기술되어 있으며, 이는 이러한 접근법을 사용하여 MPL- 및 msk-수용체들에 대한 높은 친화도 및 기능 작용적인 항체들(functional agonistic antibodies)의 단리를 기술하고 있다.
양특이성 또는 이관능의 항원 결합 단백질들
제공되는 상기 항원 결합 단백질들은 또한 앞서 기술한 바와 같은 하나 또는 그 이상의 CDRs들 또는 하나 또는 그 이상의 가변영역들을 포함하는 양특이성 및 이관능의 항체들을 포함한다. 일부 경우들에 있어서 양특이성 또는 이관능의 항체는 2개의 서로 다른 중쇄/경쇄 쌍들 및 2개의 서로 다른 결합사이트들을 갖는 인공의 잡종 항체이다. 하이브리도마들의 융합 또는 Fab' 단편들의 연결을 포함하나 이들에 제한되지 않는 다양한 방법들에 의하여 양특이성 항체들은 생산될 수 있다. 예를 들면, 송시빌라이와 라흐만의 1990, Clin. Exp. Immunol. 79:315-321; 코스텔니와 그의 동료들의 1992, J. Immunol. 148:1547-1553들을 참조하시오.
다양한 다른 형태들
제공되는 상기 항원 결합 단백질들의 일부는 앞서 기술된 상기 항원 결합 단백질들(예를 들면, 표 1 내지 표 4들 내에 기재된 상기 시퀀스들을 갖는 것들)의 변종 형태들이다. 예를 들면, 상기 항원 결합 단백질들의 일부는 표 1 내지 표 4들 내에 기재된 상기 중쇄들 또는 경쇄들, CDRs들의 가변영역들 중의 하나 또는 그 이상에서 하나 또는 그 이상의 보존적 아미노산 치환들을 갖는다.
자연적으로 발생하는 아미노산들은 공통의 측쇄 특성들에 기초하여 클래스들로 나뉘어질 수 있다:
1) 소수성 : 노르루신(norleucine), Met, Ala, Val, Leu, He;
2) 중성의 친수성 : Cys, Ser, Thr, Asn, Gln;
3) 산성 : Asp, Glu;
4) 염기성 : His, Lys, Arg;
5) 쇄의 작용에 영향을 주는 잔기들 : Gly, Pro; 및
6) 방향성 : Trp, Tyr, Phe.
보존적 아미노산 치환들에는 이들 클래스들 중의 어느 하나의 멤버의 동일한 클래스의 다른 멤버와의 교환이 포함될 수 있다. 보전적 아미노산 치환들에는 비-자연적으로 발생하는 아미노산 잔기들이 포함될 수 있으며, 이들은 전형적으로 생물학적 시스템들에서의 합성 보다는 오히려 화학적 펩티드 합성에 의해 내포되게 된다. 이들에는 펩티도미메틱(peptidomimetics) 및 다른 아미노산 부분들의 보존되거나 또는 역전된 형태들이 포함된다.
비-보존적 치환들에는 상기 클래스들 중의 어느 하나의 멤버의 다른 클래스로부터의 멤버와의 교환이 포함된다. 이러한 치환된 잔기들은 인간 항체들과 동종인 상기 항체의 영역들 내로 또는 상기 분자의 비-동종의 영역들 내로 도입될 수 있다.
이러한 교환들을 만듦에 있어, 특정의 구체예들에 따라, 아미노산들의 하이드로패스 지표(hydropathic index)가 고려될 수 있다. 단백질의 하이드로패스 프로파일은 각 아미노산에 수치적인 값("하이드로패스성 지표(hydropathy index)"을 할당하고 그리고 이들 값들을 상기 펩티드쇄를 따라 반복적으로 평균하는 것에 의해 계산된다. 각 아미노산에는 그의 소수성 및 하전 특성들에 기초하여 하이드로패스 지표가 할당된다. 이들은 다음과 같다 : 이소류신(+4.5); 발린(+4.2); 류신(+3.8); 페닐알라닌(+2.8); 시스테인/시스틴(+2.5); 메티오닌(+1.9); 알라닌(+1.8); 글리신(-0.4); 트레오닌(-0.7); 세린(-0.8); 트립토판(-0.9); 티로신(-1.3); 프롤린(-1.6); 히스티딘(-3.2); 글루타메이트(-3.5); 글루타민(-3.5); 아스파르테이트(-3.5); 아스파라긴(-3.5); 리신(-3.9); 및 아르기닌(-4.5).
단백질에 대한 상호적인 생물학적 기능을 부여하는 하이드로패스 프로파일(hydropathic profile)의 중요성은 당해 기술분야에서는 이해되고 있는 것이다(예를 들면, 카이트(Kyte)와 그의 동료들의 1982, J. MoI. Biol. 157:105-131을 참조하시오). 특정의 아미노산들이 유사한 하이드로패스 지표 또는 점수를 갖는 다른 아미노산들로 치환될 수 있으며, 여전히 유사한 생물학적 활성을 보유할 수 있다는 것이 공지되어 있다. 상기 하이드로패스 지표에 기초하여 변화들을 만듦에 있어서, 특정의 구체예들에 있어서, 그의 하이드로패스 지표들이 ±2 이내인 아미노산들의 치환이 포함된다. 일부 경우들에 있어서, 하이드로패스 지표들이 ±1 이내인 것들이 포함되며, 또한 다른 관점들에 있어서, 하이드로패스 지표들이 ±0.5 이내인 것들이 포함된다.
친수성을 기반으로 하여, 유사한 아미노산들의 치환이 효과적으로 이루어질 수 있으며, 본 경우에서와 마찬가지로 그에 의하여 생성되는 특히 생물학적으로 기능적인 단백질 또는 펩티드가 면역학적 구체예들에서 사용하기 위하여 의도된다는 것은 당해 기술분야에서는 이해될 수 있는 것이다. 특정의 구체예들에 있어서, 단백질의 가장 큰 국소적 평균 친수성은, 그의 인접하는 아미노산들의 친수성의 의해 지배되는 바와 같이, 그의 면역원성과 항원-결합 또는 상기 단백질의 생물학적 특성을 갖는 면역원성과 상관된다.
이들 아미노산 잔기들에 대하여 하기의 친수성 값들이 할당되었다: 아르기닌(+3.0); 리신(+3.0); 아스파르테이트(+3.0±l); 글루타메이트(+3.0±l); 세린(+0.3); 아스파라긴(+0.2); 글루타민(+0.2); 글리신(0); 트레오닌(-0.4); 프롤린(-0.5±1); 알라닌(-0.5); 히스티딘(-0.5); 시스테인(-1.0); 메티오닌(-1.3); 발린(-1.5); 류신(-1.8); 이소류신(-1.8); 티로신(-2.3); 페닐알라닌(-2.5) 및 트립토판(-3.4). 유사한 친수성 값들에 기초하는 변화들을 만듦에 있어서, 특정의 구체예들에 있어서, 그의 친수성 값들이 ±2 이내인 아미노산들의 치환이 포함되며, 다른 구체예들에 있어서, ±1 이내인 것들이 포함되며, 또 다른 구체예들에 있어서, ±0.5 이내인 것들이 포함된다. 일부 경우들에 있어서, 친수성의 값들에 기초하여 1차적인 아미노산 시퀀스들로부터의 에피토프들을 동정하는 것 또한 가능할 수 있다. 이들 영역들은 도한 "에피토프성 코어 영역(epitopic core regions)"으로 언급되기도 한다.
예시적인 보존적 아미노산 치환들을 표 5(보존적 아미노산 치환들)에 규정하였다.
[표 5]
숙련된 기술자는 공지의 기술들을 사용하여 본 명세서에서 규정된 바와 같은 폴리펩티드들의 적절한 변종들을 결정할 수 있을 것이다. 당해 기술분야에서 숙련된 자는 활성에 중요한 것으로 여겨지지 않는 영역들을 목표로 하는 것에 의하여 활성을 파괴함이 없이 변화될 수 있는 분자의 적절한 영역들을 동정할 수 있을 것이다. 숙련된 기술자는 또한 유사한 폴리펩티드들 중에서 보존되는 분자들의 잔기들 및 부분들을 동정할 수 있을 것이다. 다른 구체예들에 있어서, 심지어 생물학적 활성 또는 구조에 중요할 수 있는 영역들이 생물학적 활성을 파괴함이 없이 또는 상기 폴리펩티드 구조에 역으로 영향을 줌이 없이 보존적 아미노산 치환에 적용시킬 수 있다.
게다가, 당해 기술분야에서 숙련된 자는 활성 또는 구조에 중요한 유사한 폴리펩티드들 내의 잔기들을 동정하는 구조-기능 연구들을 검토할 수 있다. 이러한 비교의 관점에 있어서, 유사한 단백질들 내의 활성 또는 구조에 중요한 아미노산 잔기들에 대응하는 부분들 내의 아미노산 잔기들의 중요성을 예상할 수 있을 것이다. 당해 기술분야에서 숙련된 자는 이러한 예상되는 중요한 아미노산 잔기들에 대한 화학적으로 유사한 아미노산 치환들을 택할 수 있다.
당해 기술분야에서 숙련된 자는 또한 유사한 폴리펩티드들 내의 구조와 관련하여 3차원의 구조 및 아미노산 시퀀스를 분석할 수 있다. 이러한 정보의 관점에 있어서, 당해 기술분야에서 숙련된 자는 그의 3차원의 구종에 대한 항체의 아미노산 잔기들의 정렬을 예상할 수 있을 것이다. 당해 기술분야에서 숙련된 자는 또한 상기 단백질의 표면 상에 존재할 것으로 예상되는 아미노산 잔기들이 다른 분자들과의 중요한 상호작용들에 포함될 수 있기 때문에 이들 아미노산 잔기들에 대한 급격한 변화(radical changes)들을 만들지 않도록 선택할 수 있을 것이다. 더욱이, 당해 기술분야에서 숙련된 자는 원하는 아미노산 잔기 각각에서 단일의 아미노산 치환을 포함하는 시험 변종들을 생성할 수 있을 것이다. 계속해서 이들 변종들은 c-fms 중화 활성에 대한 분석법을 사용하여 스크리닝될 수 있으며(이하의 실시예들을 참조하시오), 그에 따라 어느 아미노산들이 변화될 수 있고 그리고 어느 아미노산들이 변화되지 않는 지에 관한 정보를 수득할 수 있을 것이다. 환언하면, 이러한 판에 박힌 실험들로부터 수집된 정보에 기초하여, 당해 기술분야에서 숙련된 자는 더 이상의 치환들이 단독으로 또는 다른 돌연변이들과의 조합으로 회피될 수 있는 아미노산 위치들을 쉽게 결정할 수 있을 것이다.
2차구조의 예상에 관한 다수의 과학출판물들이 있다. 모울트(Moult)의 1996, Curr. Op. in Biotech. 7:422-427; 추와 그의 동료들의 1974, Biochem. 13:222-245; 추와 그의 동료들의 1974, Biochemistry 113:211-222; 추와 그의 동료들의 1978, Adv. Enzymol. Relat. Areas Mol. Biol. 47:45-148; 추와 그의 동료들의 1979, Ann. Rev. Biochem. 47:251-276; 및 추와 그의 동료들의 1979, Biophys. J. 26:367-384들을 참조하시오. 더욱이, 2차구조를 예견하는 것에 대해 보조하는 컴퓨터 프로그램들이 현재 획득가능하다. 2차구조를 예상하는 하나의 방법은 동종성 모델화(homology modeling)에 기초하는 것이다. 예를 들면, 30% 이상의 시퀀스 동일성, 또는 40% 이상의 유사성을 갖는 2개의 폴리펩티드들 또는 단백질들은 유사한 구조적 위상학(structural topologies)들을 가질 수 있다. 단백질구조데이터베이스(protein structural database ; PDB)의 최근의 성장은 폴리펩티드들 또는 단백질들의 구조 내의 겹(folds)들의 잠재적인 수를 포함하여 2차구조의 증가된 예견성을 제공한다. 홀름과 그의 동료들의 1999, Nucl. Acid. Res. 27:244-247를 참조하시오. 주어진 폴리펩티드 또는 단백질 내에 제한된 수의 겹들이 존재하고 그리고 일단 구조들의 절대값(critical number)이 해결되면 구조적인 예상이 극적으로 보다 정확해질 수 있다는 것이 제안되었다(브레너(Brenner)와 그의 동료들의 1997, Curr. Op. Struct. Biol. 7:369-376).
2차구조를 예상하는 별도의 방법들에는 "스레딩(threading)"(존스(Jones)의 1997, Curr. Opin. Struct. Biol. 7:377-387; 시플(Sippl)과 그의 동료들의 1996, Structure 4:15-19)), "프로파일 분석(profile analysis)" (보위(Bowie)와 그의 동료들의 1991, Science 253:164-170; 그립스코프(Gribskov)와 그의 동료들의 1990, Meth. Enzym. 183:146-159; 그립스코프와 그의 동료들의 1987, Proc. Nat. Acad. Sci. 84:4355-4358), 및 "혁신적 연결(evolutionary linkage)"(홀름의 위의 1999 문헌; 및 브렌너(Brenner)의 위의 1997 문헌들을 참조)들이 포함된다.
일부 구체예들에 있어서, 아미노산 치환들은 (1) 단백질 가수분해(proteolysis)에 대한 감수성을 감소, (2) 산화에 대한 감수성을 감소, (3) 단백질 착체(protein complexes)들의 형성에 대한 결합 친화도를 변경, (4) 리간드 또는 항원 결합 친화도를 변경 및/또는 (4) 이러한 폴리펩티드들에 대한 다른 물리화학적 또는 기능적인 특성들을 부여 또는 변형시킨다. 예를 들면, 단일 또는 다중의 아미노산 치환들(특정의 구체예들에 있어서는, 보존적 아미노산 치환들)이 자연적으로 발생하는 시퀀스 내에서 이루어질 수 있다. 치환들은 분자간 접촉들을 형성하는 상기 도메인(들)의 외측에 위치하는 상기 항체의 부분 내에서 이루어질 수 있다. 이러한 구체예들에 있어서, 대체로 부모 시퀀스(예를 들면, 부모 또는 자연의 항원 결합 단백질을 특징하는 상기 2차구조를 방해하지 않는 하나 또는 그 이상의 대체 아미노산들)의 구조적 특성들을 변화시키지 않는 보존적 아미노산 치환들이 사용될 수 있다. 기술분야에서 인식된 폴리펩티드 2차 및 3차구조들의 예들이 단백질들, 구조들 및 분자상 원리들(Structures and Molecular Principles ; (Creighton, Ed.), 1984, W. H. New York: Freeman and Company); 단백질 구조의 입문(Introduction to Protein Structure ; (Branden and Tooze, eds.), 1991, New York: Garland Publishing); 및 소른톤(Thornton)과 그의 동료들의 1991, Nature 354:105들에서 기술되어 있으며, 이들을 각각 본 명세서에서 참조로 인용한다.
별도의 바람직한 항체 변종들에는 시스테인 변종들이 포함되며, 여기에서 부모 또는 자연의 아미노산 시퀀스 내의 하나 또는 그 이상의 시스테인 잔기들이 탈락되거나 또는 다른 아미노산(예를 들면, 세린)으로 치환된다. 시스테인 변종들은 유용하며 그 중에서도 항체들이 생물학적으로 활성인 형태로 다시 접혀져야만 하는 경우에 유용하다. 시스테인 변종들은 자연의 항체들 보다 보다 적은 시스테인 잔기들을 가질 수 있으며, 전형적으로 짝지워지지 않은 시스테인들로부터 야기되는 상호작용들을 최소화하기 위하여 짝수를 갖는다.
기술되는 상기 중쇄 및 경쇄들, 가변영역들, 도메인들 및 CDRs들은 c-fms 폴리펩티드에 특이적으로 결합할 수 있는 항원 결합 영역을 포함하는 폴리펩티드들을 준비하는 데 사용될 수 있다. 예를 들면, 표 3 및 표 4에 기재된 상기 CDRs들 중의 하나 또는 그 이상이 분자(예를 들면, 폴리펩티드) 내로 공유적으로 또는 비공유적으로 내포되어 면역접착을 형성할 수 있다. 면역접착은 보다 큰 폴리펩티드쇄들의 일부로 상기 CDR(s)들을 내포시키거나, 상기 CDR(s)들을 다른 폴리펩티드쇄에 공유적으로 연결시키거나 또는 상기 CDR(s)들을 비공유적으로 내포시킬 수 있다. 상기 CDR(s)들은 대상의 특정의 항원(예를 들면, c-fms 폴리펩티드 또는 그들의 에피토프)에 특이적으로 결합하는 면역접착을 가능하게 한다.
상기 가변영역 도메인들에 기초하는 모방체들(예를 들면, "펩티드 모방체들(peptide mimetics)" 또는 "펩티도미메틱들") 및 본 명세서에서 기술되는 CDRs들이 또한 제공된다. 이들 유사체들은 펩티드, 비-펩티드 또는 펩티드와 비-펩티드 영역들의 조합이 될 수 있다. 모든 목적들에 대하여 참조로 본 명세서에서 인용되는 파우셔어(Fauchere)의 1986, Adv. Drug Res. 15:29; 베버와 프라이딩거(Veber and Freidinger)의 1985, TINS p. 392; 및 에반스와 그의 동료들의 1987, J. Med. Chem. 30:1229들을 참조하시오. 치료적으로 유용한 펩티드들에 구조적으로 유사한 펩티드 모방체들이 유사한 치료적 또는 예방적인 효과들을 생성하는 데 사용될 수 있다. 이러한 화합물들은 종종 컴퓨터화된 분자 모델링(computerized modlecular modeling)의 도움으로 개발된다. 대체로, 펩티도미메틱들은 본 명세서에서는 c-fms에 특이적으로 결합하는 능력 등과 같은 원하는 생물학적 활성을 나타내는 항체에 구조적으로 유사하나 그러나 당해 기술분야에서 공지된 방법들에 의하여 -CH2NH-, -CH2S-, -CH2-CH2-, -CH-CH-(시스 및 트랜스), - COCH2-, -CH(OH)CH2- 및 -CH2SO- 들로부터 선택되는 연결에 의해 선택적으로 대체되는 하나 또는 그 이상의 펩티드 연결들을 갖는 단백질들이다. 동일한 형태의 D-아미노산(예를 들면, L-리신을 대신하는 D-리신)에 대한 합치 시퀀스의 하나 또는 그 이상의 아미노산들의 체계적인 치환은 특정의 구체예들에서 사용되어 보다 안정한 단백질들을 생성할 수 있다. 게다가, 합치 시퀀스 또는 대체로 동일한 합치 시퀀스 변이를 포함하는 제한된 펩티드들이 당해 기술분야에서 공지된 방법들, 예를 들면, 상기 펩티드를 고리화(cyclize)하는 분자내 이황화 브릿지(intramolecular disulfide bridges)들을 형성할 수 있는 내부 시스테인 잔기들을 첨가하는 것에 의하여 생산될 수 있다(여기에서 참조로 인용되는 리조와 기어라쉬(Rizo and Gierasch)의 1992, Ann. Rev. Biochem. 61:387).
본 명세서에서 기술되는 상기 항원 결합 단백질들의 유도체들이 또한 제공된다. 유도화된 항원 결합 단백질들은 상기 항체 또는 단편에 특정의 용도에서의 증가된 반감기 등과 같은 원하는 특성을 부여하는 임의의 분자 또는 물질을 포함할 수 있다. 상기 유도화된 항원 결합 단백질에는, 예를 들면, 검출가능한(또는 표지된) 부분(예를 들면, 방사성, 비색성(colorimetric), 항원성(antigenic) 또는 효소분자, 검출가능한 비드(bead)(자성비드 또는 전자밀집(예를 들면, 금)비드), 또는 다른 분자에 결합하는 분자(예를 들면, 비오틴 또는 스트렙타비딘(sterptavidin)), 치료적 또는 진단적 부분(예를 들면, 방사성, 세포독성, 또는 약제학적으로 활성인 부분), 또는 특정의 용도(예를 들면, 인간 대상체 등과 같은 대상체에의 투여, 또는 다른 생체내 또는 시험관내 용도들)에 대한 상기 항원 결합 단백질의 적합성을 증가시키는 분자들이 포함될 수 있다. 항원 결합 단백질의을 유도화 하는 데 사용될 수 있는 분자들의 예들에는 알부민(예를 들면, 인간 혈청 알부민) 및 폴리에틸렌글리콜(PEG)들이 포함된다. 항원 결합 단백질들의 알부민-연결된 그리고 피이지화된 유도체들은 당해 기술분야에서 공지된 기술들을 사용하여 제조될 수 있다. 특정의 항원 결합 단백질들은 본 명세서에서 기술되는 바와 같은 피이지화된 단일쇄 폴리펩티드들을 포함한다. 하나의 구체예에 있어서, 상기 항원 결합 단백질은 티록신결합전알부민(TTR ; transthyretin) 또는 TTR 변종에 콘쥬게이트 되거나 또는 달리 연결된다. 상기 TTR 또는 TTR 변종은, 예를 들면, 덱스트란(dextran), 폴리(n-비닐피롤리돈)(poly(n-vinyl pyrrolidone)), 폴리에틸렌글리콜, 폴리프로필렌글리콜 호모중합체, 폴리프로필렌옥사이드/에틸렌옥사이드 공중합체, 폴리옥시에틸렌화폴리올 및 폴리비닐알코올들로 이루어지는 그룹으로부터 선택되는 화학약품으로 화학적으로 변형될 수 있다.
다른 유도체들에는 c-fms 항원 결합 단백질의 N-말단 또는 C-말단에 융합되는 이종기원의 폴리펩티드들을 포함하는 재조합 융합 단백질들의 발현에 의하는 것과 같은 c-fms 항원 결합 단백질들의 다른 단백질들 또는 폴리펩티드들과의 공유적 또는 집합적 콘쥬게이트들(aggregative conjugates)이 포함된다. 예를 들면, 상기 콘쥬게이트된 펩티드는 이종기원의 신호(또는 리더) 폴리펩티드, 예를 들면, 이스트 알파-인자 리더(yeast alpha-factor leader) 또는 에피토프 태그 등과 같은 펩티드가 될 수 있다. c-fms 항원 결합 단백질-함유 융합 단백질들에는 상기 c-fms 항원 결합 단백질의 정제 및 동정을 용이하게 하도록 첨가되는 펩티드들이 포함될 수 있다(예를 들면, 폴리-His). c-fms 항원 결합 단백질은 또한 호프(Hopp)와 그의 동료들의 1988, Bio/Technology 6:1204; 및 미합중국 특허 제5,011,912호들에서 기술된 바와 같은 FLAG 펩티드에 연결될 수 있다. 상기 FLAG 펩티드는 고도로 항원성이며 또한 특이적인 모노클로날 항체(mAb)에 의하여 가역적으로 결합하는 에피토프를 제공하여 발현된 재조합 단백질의 신속한 분석 및 용이한 정제를 가능하게 한다. 상기 FLAG 펩티드가 주어진 폴리펩티드에 융합된 융합 단백질들을 제조하는 데 유용한 시약들은 상용적으로 획득가능하다(시그마사, 미합중국 미주리주 세인트루이스시 소재).
하나 또는 그 이상의 c-fms 항원 결합 단백질들을 포함하는 올리고머들이 c-fms 길항제들로 사용될 수 있다. 올리고머들은 공유적으로-연결되거나 또는 비-공유적으로-연결되는 이량체(dimers), 삼량체(trimers) 또는 보다 높은 올리고머들의 형태가 될 수 있다. 둘 또는 그 이상의 c-fms 항원 결합 단백질들을 포함하는 올리고머들이 호모이량체(homodimer)인 하나의 구체예에 대하여 사용된느 것이 고려된다. 다른 올리고머들에는 헤테로이량체(heterodimers), 호모삼량체(homotrimers), 헤테로삼량체(heterotrimers), 호모사량체(homotetramers), 헤테로사량체(heterotetramers) 등이 포함된다.
하나의 구체예는 상기 c-fms 항원 결합 단백질들에 융합된 펩티드 부분들 사이에서의 공유적 또는 비-공유적 상호작용들을 통하여 결합되는 다중의 c-fms-결합 폴리펩티드들을 포함하는 올리고머들에 지향된다. 이러한 펩티드들에는 펩티드 링커(peptide linkers(스페이서(spacers)), 또는 올리고머화(oligomerization)를 촉진하는 특성을 갖는 펩티드들이 포함될 수 있다. 이하에서 보다 상세하게 기술되는 바와 같이, c-fms 항원 결합 단백질들에 부착되어 그의 올리고머화를 촉진시킬 수 있는 펩티드들 중에서도 류신 지퍼(leucine zippers) 및 항체들로부터 유도되는 특정의 폴리펩티드들이 있다.
특정의 구체예들에 있어서, 상기 올리고머들은 2 내지 4개의 c-fms 항원 결합 단백질들을 포함한다. 상기 올리고머의 상기 c-fms 항원 결합 단백질 부분들은 앞서 기술된 임의의 형태들, 예를 들면, 변종들 또는 단편들이 될 수 있다. 바람직하게는, 상기 올리고머는 c-fms 결합 활성을 갖는 c-fms 항원 결합 단백질들을 포함한다.
하나의 구체예에 있어서, 올리고머는 면역글로블린들로부터 유도되는 폴리펩티드들을 이용하여 제조된다. 항체-유도 폴리펩티드들(상기 Fc 도메인을 포함하여)의 여러 부분들에 융합되는 특정의 이종기원의 폴리펩티드들을 포함하는 융합 단백질들의 제조는, 예를 들면, 아쉬케나지(Ashkenazi)와 그의 동료들의 1991, Proc. Natl. Acad. Sci. USA 88:10535; 바이른(Byrn)과 그의 동료들의 1990, Nature 344:677; 및 홀렌바우(Hollenbaugh)와 그의 동료들의 1992 "Construction of Immunoglobulin Fusion Proteins", in Current Protocols in Immunology, Suppl. 4, pages 10.19.1-10.19.11들에 의해 기술되었다.
하나의 구체예는 c-fms 항원 결합 단백질을 항체의 상기 Fc 영역에 융합시키는 것에 의해 생성되는 2개의 융합 단백질들을 포함하는 이량체로 지향된다. 상기 이량체는, 예를 들면, 상기 융합 단백질을 인코딩하는 유전자 융합물을 적절한 발현 벡터 내로 삽입시키고, 상기 재조합 발현 벡터로 형질전환된 숙주세포들 내의 유전자 융합물을 발현시키고, 그리고 상기 발현된 융합 단백질을 항체 분자들과 마찬가지로 수집하도록 하고, 그에 의하여 상기 Fc 부분들 사이에 쇄간의 이황하 결합들이 형성되어 상기 이량체를 수득하도록 하는 것에 의하여 제조될 수 있다.
본 명세서에서 사용되는 바와 같은 용어 "Fc 폴리펩티드"는 항체의 상기 Fc 영역으로부터 유도되는 폴리펩티드들의 자연의 및 돌연변이단백질 형태들을 포함한다. 이량체화(dimerization)를 촉진하는 힌지영역(hinge region)을 포함하는 이러한 폴리펩티드들의 끝이 절단된 형태들이 또한 포함된다. Fc 부분들(및 그들로부터 형성되는 올리고머들)을 포함하는 융합 단백질들은 단백질 A 또는 단백질 G 컬럼들에 대한 친화도 크로마토그래피에 의한 용이한 정제의 잇점을 제공한다.
국제특허공개공보 제WO 93/10151호 및 미합중국 특허 제5,426,048호와 동 제5,262,522호들에서 기술된 하나의 적절한 Fc 폴리펩티드는 인간 IgG1 항체의 상기 Fc 영역의 N-말단 힌지영역으로부터 자연의 C-말단까지 연장되는 단일쇄 폴리펩티드이다. 다른 유용한 Fc 폴리펩티드는 미합중국 특허 제5,457,035호 및 바움(Baum)과 그의 동료들의 1994, EMBO J. 13:3992-4001에서 기술된 Fc 돌연변이단백질이다. 이 돌연변이단백질의 아미노산 시퀀스는 아미노산 19가 Leu에서 Ala로 바뀌고 그리고 아미노산 20이 Leu로부터 Glu로 바뀌고 그리고 아미노산 22가 Gly로부터 Ala로 바뀐 것을 제외하고는 국제특허공개공보 제WO 93/10151호에서 제공되는 자연의 Fc 시퀀스의 그것과 동일하다. 상기 돌연변이단백질은 Fc 수용체들에 대하여 감소된 친화도를 나타낸다.
다른 구체예들에 있어서, 본 명세서에서 기술된 바와 같은 c-fms 항원 결합 단백질의 상기 중쇄 및/또는 경쇄들의 상기 가변영역은 항체 중쇄 및/또는 경쇄의 가변영역에 대하여 치환될 수 있다.
달리, 상기 올리고머는 펩티드 링커(스페이서 펩티드들)과 함께 또는 그것들 없이 다중의 c-fms 항원 결합 단백질들을 포함하는 융합 단백질이다. 적절한 펩티드 링커들 중에서는 미합중국 특허 제4,751,180호 및 동 제4,935,233호에서 기술된 것들이 있다.
올리고머성의 c-fms 항원 결합 단백질 유도체들을 제조하기 위한 다른 방법들에는 류신 지퍼의 사용이 포함된다. 류신 지퍼 도메인들은 그들이 발견되는 상기 단백질들의 올리고머화를 촉진하는 펩티드들이다. 류신 지퍼들은 원래 여러 DNA-결합 단백질들에서 동정되었으며(랜드슐츠(Landschulz)와 그의 동료들의 1988, Science 240:1759), 그때부터 다양한 서로 다른 단백질들 내에서 발견되었다. 공지된 류신 지퍼들 중에는 자연적으로 발생하는 펩티드들 및 이량체하거나(dimerize) 또는 삼량체화(trimerize)하는 그들의 유도체들이 있다. 가용성의 올리고머성 단백질들을 생산하기에 적절한 류신 지퍼 도메인들의 예들은 국제특허공개공보 제WO 94/10308호에 기술되어 있으며, 또한 폐의 계면 단백질 D(lung surfactant protein D)(SPD)로부터 유도되는 류신 지퍼가 호페(Hoppe)와 그의 동료들의 1994, FEBS Letters 344:191에 기술되어 있으며, 자에 참조로 인용한다. 그에 융합되는 이종기원의 단백질의 안정한 삼량체화를 허용하는 변형된 류신지퍼의 사용이 팬스로우(Fanslow)와 그의 동료들의 1994, Semin. Immunol. 6:267-278에 기술되어 있다. 하나의 접근법에 있어서, c-fms 항원 결합 단백질 단편 또는 류신 지퍼 펩티드에 융합된 유도체를 포함하는 재조합 융합 단백질들이 적절한 숙주세포들 내에서 발현되고, 그리고 상기 가용성 올리고머성 c-fms 항원 결합 단백질 단편들 또는 이를 형성하는 유도체들은 그 배양 상청액으로부터 회수된다.
제공되는 일부 항원 결합 단백질들은, 예를 들어, 이하의 실시예들에서 기술된 바에 따라 측정된, c-fms에 대하여 적어도 104/M*초, 적어도 105/M*초, 적어도 106/M*초의 온-레이트(on-rate ; 해리상수)(ka)를 갖는다. 제공되는 특정의 항원 결합 단백질들은 낮은 해리 속도(dissociation rate) 또는 오프-레이트(off-rate)를 갖는다. 예를 들면, 일부 항원 결합 단백질들은 1*10-2s-1 또는 1*10-3s-1 또는 1*10-4s-1 또는 1*10-5s-1의 kD(오프-레이트)를 갖는다. 특정의 구체예들에 있어서, 상기 항원 결합 단백질은 25pM, 50pM, 100pM, 500pM, 1nM, 5nM, 10nM, 25nM 또는 50 nM 이하의 KD(평행 결합 친화도 ; equilibrium binding affinity)을 갖는다.
다른 관점은 시험관 내 또는 생체 내(예를 들면, 인간 대상체에 투여되는 경우)에서 적어도 하루의 반감기를 갖는 항원-결합 단백질을 제공한다. 하나의 구체예에 있어서, 상기 항원 결합 단백질은 적어도 3일의 반감기를 갖는다. 다른 구체예에 있어서, 상기 항체 또는 그들의 부분은 4일 또는 그 이상의 반감기를 갖는다. 또 다른 구체예에 있어서, 상기 항체 또는 그들의 부분은 8일 또는 그 이상의 반감기를 갖는다. 또 다른 구체예에 있어서, 상기 항체 또는 그들의 항원-결합 부분은 이것이 유도체화되지 않거나 또는 변형되지 않은 항체에 비하여 보다 긴 반감기를 갖도록 유도체화되거나 또는 변형된다. 또 다른 구체예에 있어서, 상기 항원 결합 단백질은 여기에 참고로 인용되는 2000년 2월 24일 공개된 국제특허공개공보 제WO 00/09560호에 기술된 바와 같은 혈청 반감기를 증가시키는 점 돌연변이(point mutations)들을 포함한다.
글리코실화(Glycosylation)
상기 항원-결합 단백질은 자연의 종들에서 발견되는 것과 다르거나 또는 그로부터 변경되는 글리코실화 패턴(glycosylation pattern)을 가질 수 있다. 당해 기술분야에서 공지된 바와 같이, 글리코실화 패턴들은 상기 단백질의 시퀀스(예를 들면, 이하에서 기술되는 특정의 글리코실화 아미노산 잔기들의 존재 또는 부재) 또는 상기 단백질이 생산되는 숙주세포 또는 유기체 둘 다에 의존적일 수 있다. 특정의 발현 시스템들이 이하에서 기술된다.
폴리펩티드들의 글리코실화는 전형적으로는 N-연결 또는 O-연결이다. N-연결은 아스파라긴 잔기의 측쇄의 탄수화물 부분의 부착을 의미한다. X가 프롤린을 제외한 임의의 아미노산인 트리-펩티드(tri-peptide) 시퀀스들인 아스파라긴-X-세린 및 아스파라긴-X-트레오닌들은 상기 아스파라긴 측쇄에의 상기 탄수화물 부분의 효소부착(enzymatic attachment)에 대한 인식 시퀀스(recognition sequences)들이다. 따라서, 폴리펩티드 내의 이들 트리-펩티드 시퀀스들 둘 중의 어느 것의 존재는 잠재적인 글리코실화 사이트(glycosylation site)를 생성한다. 비록 5-히드록시프롤린 또는 5-히드록시리신들이 또한 사용될 수 있기는 하나, O-연결 글리코실화는 당 N-아세틸갈락토사민, 갈락토스 또는 자일로스들 중의 하나의 히드록시아미노산, 가장 통상적으로는 세린 또는 트레오닌에의 부착을 의미한다.
상기 항원 결합 단백질에의 글리코실화 사이트들의 부가는 상기 아미노산 시퀀스가 하나 또는 그 이상의 앞서 기술된 트리-펩티드 시퀀스(N-연결 글리코실화 사이트들에 대하여)를 포함하도록 상기 아미노산 시퀀스를 변형시키는 것에 의해 편리하게 수행된다. 상기 변형은 또한 개시 시퀀스(starting sequence)(O-연결 글리코실화 사이트들에 대하여)에 하나 또는 그 이상의 세린 또는 트레오닌 잔기들의 첨가 또는 치환에 의해 이루어질 수 있다. 편의를 위하여, 상기 항원 결합 단백질 아미노산 시퀀스는 DNA 수준에서의 변화를 통하여, 특히 사전선택된 염기들에서의 목표 폴리펩티드를 인코딩하는 상기 DNA를 돌연변이시켜 원하는 아미노산들로 번역될 수 있는 코돈(codons)들이 생성되도록 하는 것에 의하여 변경될 수 있다.
상기 항원 결합 단백질 상의 탄수화물 부분들의 수를 증가시키는 다른 수단은 글리코시드들의 상기 단백질에의 화학커플링 또는 효소커플링(enzymatic coupling)에 의하는 것이다. 이들 절차들은 이들이 N- 및 O-연결 글리코실화를 위한 글리코실화 능력들을 갖는 숙주세포 내에서의 상기 단백질의 생산을 요구하지 않는 다는 점에서 유리하다. 사용된 커플링 모드(coupling mode)에 따라, 상기 당(들)은 (a) 아르기닌 및 히스티딘, (b) 유리 카르복실기들, (c) 시스테인의 그것 등과 같은 유리 설피드릴기들, (d) 세린, 트레오닌 또는 히드록시프롤린의 그것 등과 같은 유리 히드록실기들, (e) 페닐알라닌, 티로신 또는 트립토판의 그것 등과 같은 방향족 잔기들 또는 (f) 글루타민의 아미노기들에 부착될 수 있다. 이들 방법들은 1987년 9월 11일 공개된 국제특허공개공보 제WO 87/05330호 및 아플린과 리스톤(Aplin and Wriston)의 1981, CRC Crit. Rev, Biochem., pp. 259-306에서 기술된다.
상기 개시 항원 결합 단백질 상에 존재하는 탄수화물 부분들의 제거는 화학적으로 또는 효소적으로 수행될 수 있다. 화학적 탈글리코실화(deglycosylation)는 상기 단백질의 트리플루오로메탄설폰산 화합물 또는 등가의 화합물에의 노출을 요구한다. 이러한 처치는 연결당(linking sugar ; N-아세틸글루코사민 또는 N-아세틸갈락토사민)을 제외한 대부분 또는 전체 당들의 개열이라는 결과를 가져오는 한편으로 상기 폴리펩티드는 손상되지 않은 채로 남긴다. 화학적 탈글리코실화는 하키무딘(Hakimuddin)과 그의 동료들의 1987, Arch. Biochem. Biophys. 259:52에 의해 그리고 엣지(Edge)와 그의 동료들의 1981, Anal. Biochem. 118:131에 의해 기술된다. 폴리펩티드들 상의 탄수화물 부분들의 효소개열은 토타쿠라(Thotakura)와 그의 동료들의 1987, Meth. Enzymol. 138:350에 의해 기술된 바와 같은 다양한 엔도-글리코시다아제 및 엑소-글리코시다아제들의 사용에 의하여 수행될 수 있다. 잠재적인 글리코실화 사이트들에서의 글리코실화는 더스킨(Duskin)과 그의 동료들의 1982, J. Biol. Chem. 257:3105에 의해 기술된 바와 같은 투니카마이신(tunicamycin)의 사용에 의해 방지될 수 있다. 투니카마이신은 단백질-N-글리코시드 연결들의 형성을 차단한다.
따라서, 상기 항원 결합 단백질들의 글리코실화 변형들이 관점들에 포함되며, 여기에서 글리코실화 사이트(들)의 개수 및/또는 형태는 부모 폴리펩티드의 상기 아미노산 시퀀스들과 비교하여 변형된다. 특정의 구체예들에 있어서, 항체 단백질 변형들에는 자연의 항체에 비해 보다 크거나 또는 보다 작은 수의 N-연결 글리코실화 사이트들이 포함된다. N-연결 글리코실화 사이트는 시퀀스 Asn-X-Ser 또는 Asn-X-Thr에 의해 특징지워지며, 여기에서 X로 표시된 상기 아미노산 잔기는 프롤린을 제외한 임의의 아미노산 잔기가 될 수 있다. 이러한 시퀀스를 생성하기 위한 아미노산 잔기들의 치환은 N-연결 탄수화물쇄의 첨가에 대한 잠재적인 새로운 사이트를 제공한다. 달리, 이러한 시퀀스를 제거하거나 또는 변형시키는 치환들은 자연의 폴리펩티드 내에 존재하는 N-연결 탄수화물쇄의 첨가를 방지할 것이다. 예를 들면, 상기 글리코실화는 Asn의 탈락에 의하거나 또는 상기 Asn의 다른 아미노산으로의 치환에 의해 감소될 수 있다. 다른 구체예들에 있어서, 하나 또는 그 이상의 새로운 N-연결 사이트들이 생성된다. 항체들은 전형적으로 Fc 영역 내에 N-연결 글리코실화 사이트를 갖는다.
표지들 및 효과기들(Labels and Effector Groups)
일부 구체예들에 있어서, 상기 항원-결합은 하나 또는 그 이상의 표지들을 포함한다. 용어 "표지화기(labeling group)" 또는 "표지(label)"은 임의의 검출가능한 표지를 의미한다. 적절한 표지화기들의 예들에는 방사성 동위원소들 또는 방사성 핵종들(예를 들면, 3H, 14C, 15N, 35S, 90Y, 99Tc, 111In, 125I, 131I), 형광기들(예를 들면, FITC, 로다민, 란탄계열 인광물질), 효소기들(enzymatic groups ; 예를 들면, 고추냉이과산화효소(horseradish peroxidase), 베타-갈락토시다아제(β-galactosidase), 루시퍼라아제(luciferase), 알칼리포스파타아제(alkaline phosphatase)), 화학발광기들(chemiluminescent groups), 비오티닐기들(biotinyl groups) 또는 2차 리포터(secondary reporter ; 예를 들면, 루신 지퍼 쌍 시퀀스들, 2차 항체들에 대한 바인딩 사이트들, 금속 바인딩 도메인들, 에피토프 태그들)에 의해 인식되는 사전설정된 폴리펩티드 에피토프들이 포함되나, 이들에 제한되는 것은 아니다. 일부 구체예들에 있어서, 상기 표지화기는 잠재적인 입체장애를 감소시키기 위한 다양한 길이들의 스페이서 아암(spacer arms)들을 통하여 상기 항원 결합 단백질에 결합된다. 단백질들을 표지화하기 위한 다양한 방법들이 당해 기술분야에서 공지되어 있으며, 맞는 것으로 보여지는 바에 따라 사용될 수 있다.
용어 "효과기(effector group)"는 세포독성제로서 작용하는 항원 결합 단백질에 결합되는 임의의 기를 의미한다.적절한 효과기들에 대한 예들은 방사성 동위원소 또는 방사성 핵종(예를 들면, 3H, 14C, 15N, 35S, 90Y, 99Tc, 111In, 125I, 131I)들이다. 다른 적절한 기들에는 톡신, 치료적인 기들 또는 화학요법적인 기들이 포함된다. 적절한 기들의 예들에는 칼리케아미신(calicheamicin), 아우리스타틴(auristatin), 겔다나마이신(geldanamycin) 및 메이탄신(maytansine)들이 포함된다. 일부 구체예들에 있어서, 상기 효과기는 잠재적인 입체장애를 감소시키기 위한 다양한 길이들의 스페이서 아암들을 통하여 상기 항원 결합 단백질에 결합된다.
대체로, 표지들은 그들이 검출되어야 할 분석법에 따라 a) 방사성 동위원소 또는 중동위원소(heavy isotopes)들일 수 있는 동위원소 표지들; b) 자성표지들(예를 들면, 자성입자들); c) 레독스 활성 부분들(redox active moieties); d) 광학염료들; 효소기들(예를 들면, 고추냉이과산화효소, 베타-갈락토시다아제, 루시퍼라아제, 알칼리포스파타아제); e) 비오티닐화기들; 및 f) 2차 리포에 의해 인식되는 사전설정된 폴리펩티드 에피토프 태그들 등의 다양한 클래스들 내에 속하게 된다. 일부 구체예들에 있어서, 상기 표지화기는 잠재적인 입체장애를 감소시키기 위한 다양한 길이들의 스페이서 아암들을 통하여 상기 항원 결합 단백질에 결합된다. 단백질들을 표지화하기 위한 여러 방법들이 당해 기술분야에서 공지되어 있다.
특정의 표지들에는 발색단(chromophores), 인광물질들 및 형광물질들을 포함하나 이들에 제한되지 않는 광학염료들이 포함되며, 후자는 많은 경우들에서 특이적이다. 형광물질들은 "작은 분자" 형광체(fluores) 또는 단백질형광체가 될 수 있다.
"형광 표지(fluorescent label)"에 대하여는 이는 그의 고유의 형광특성들을 통하여 검출될 수 있는 임의의 분자를 의미한다. 적절한 형광 표지들에는 플루오레세인(fluorescein), 로다민, 테트라메틸로다민(tetramethylrhodamine), 에오신(eosin), 에리트로신(erythrosin), 쿠마린(coumarin), 메틸-쿠마린(methyl-coumarins), 피렌(pyrene), 말라사이트그린(Malacite green), 스틸벤(stilbene), 루시퍼옐로우(Lucifer Yellow)), 케스케이드블루제이(Cascade BlueJ), 텍사스레드(Texas Red), 이에단스(IAEDANS), 에단스(EDANS), 보디파이에프엘(BODIPY FL), 엘씨레드640(LC Red 640), 씨와이5(Cy 5), 씨와이5.5, 엘씨레드705, 오레곤그린(Oregon green), 알렉사-플루오르염료들(알렉사플루오르350(Alexa Fluor 350), 알렉사플루오르430, 알렉사플루오르488, 알렉사플루오르546, 알렉사플루오르568, 알렉사플루오르594, 알렉사플루오르633, 알렉사플루오르660, 알렉사플루오르680), 케스케이드블루, 케스케이드옐로우와 알-피코에리트린(R-phycoerythrin ; PE ; 미합중국 오레곤주 유진 소재의 몰레큘라프로브스(Molecular Probes)사 제품), FITC, 로다민 및 텍사스레드(미합중국 일리노이주 록포드 소재의 피어스(Pierce)사 제품), 씨와이5, 씨와이5.5, 씨와이7(미합중국 펜실베니어주 피츠버그 소재의 어메리칸 라이프 사이언스(Amersham Life Science)사 제품)들이 포함되나, 이들에 제한되는 것은 아니다. 형광물질들을 포함하는 적절한 광학염료들은 리차드 피. 호그랜드(Richard P. Haugland)의 분자탐침 핸드북(MOLECULAR PROBES HANDBOOK)에 기술되어 있으며, 여기에서 이를 특별히 참조로 인용한다.
적절한 단백질형광체 표지들에는 또한 레닐라(Renilla), 프틸로사르쿠스(Ptilosarcus) 또는 GFP(찰피에(Chalfie)와 그의 동료들의 1994, Science 263:802-805)의 아에쿠오레아종(Aequorea species), EGFP(클론테크 랩스 인코포레이티드(Clontech Labs., Inc.), 유전자은행 기탁번호 유55762(Genbank Accession Number U55762))들을 포함하는 녹색형광단백질(green fluorescent protein), 청색형광단백질(BFP, 캐나다 퀘벡 소재의 퀀텀 바이오테크놀로지스 인코포레이티드(Quantum Biotechnologies, Inc.); 스타우버(Stauber)의 1998, Biotechniques 24:462-471 ; 하임(Heim)과 그의 동료들의 1996, Curr. Biol. 6:178-182), 강화황색형광단백질(EYFP, 클론테크 랩스 인코포레이티드), 루시퍼라아제(이치키(Ichiki)와 그의 동료들의 1993, J. Immunol. 150:5408-5417), 베타 갈락토시다아제(노란(Nolan)과 그의 동료들의 1988, Proc. Natl Acad. Sci. U.S.A. 85:2603-2607) 및 레닐라(Renilla ; 국제특허공개공보 제WO92/15673호, 동 제WO95/07463호, 동 제WO98/14605호, 동 제WO98/26277호, 동 제WO99/49019호, 미합중국 특허 제5292658호, 동 제5418155호, 동 제5683888호, 동 제5741668호, 동 제5777079호, 동 제5804387호, 동 제5874304호, 동 제5876995호, 동 제5925558호)들이 포함되나, 이들에 제한되는 것은 아니다.
C-fms 항원 결합 단백질들을 인코딩하는 핵산들
항체의 두 쇄들 중의 하나를 인코딩하는 핵산들 또는 단편, 유도체, 돌연변이 단백질 또는 그들의 변종, 중쇄 가변영역들 또는 단지 CDRs들을 인코딩하는 폴리뉴클레오티드, 하이브리드화 탐침(hybridization probes)들로서 사용하기에 충분한 폴리뉴클레오티드, 폴리펩티드를 인코딩하는 폴리뉴클레오티드를 동정, 분석, 돌연변이화 또는 증폭을 위한 PCR 프라이머 또는 시퀀싱 프라이머, 폴리뉴클레오티드의 발현을 저해하기 위한 항-감작 핵산 및 앞서의 것들의 상보적 시퀀스들을 포함하여 본 명세서에서 기술되는 상기 항원 결합 단백질들을 인코딩하는 핵산 또는 그들의 부분들이 또한 제공된다. 상기 핵산들은 임의의 길이가 될 수 있다. 예를 들면, 이들은 길이에 있어서 5, 10, 15, 20, 25, 30, 35, 40, 45, 50, 75, 100, 125, 150, 175, 200, 250, 300, 350, 400, 450, 500, 750, 1,000, 1,500, 3,000, 5,000 또는 그 이상의 뉴클레오티드들이 될 수 있거나 및/또는 하나 또는 그 이상의 부가의 시퀀스들, 예를 들면, 조절시퀀스(regulatory sequences)들을 포함하거나 및/또는 보다 큰 핵산들, 예를 들면, 벡터의 일부가 될 수 있다. 상기 핵산들은 단일-가닥 또는 이중-가닥이 될 수 있으며, RNA 및/또는 DNA 뉴클레오티드들 및 이들의 인공의 변종들(예를 들면, 펩티드핵산(peptide nucleic acids))들을 포함할 수 있다. 표 6(중쇄 및 경쇄 불변영역들을 인코딩하는 예시적인 핵산 시퀀스들)은 IgG2 중쇄 불변영역 및 IgG2 카파 경쇄 불변영역을 인코딩하는 예시적인 핵산 시퀀스들을 나타내고 있다. 본 명세서에서 제공되는 임의의 가변영역은 이들 불변영역들에 부착되어 완전한 중쇄 및 경쇄 시퀀스들을 형성할 수 있다. 그러나, 이들 불변영역 시퀀스들은 단지 특정의 예들로서 제공되는 것임은 이해되어야 한다. 일부 구체예들에 있어서, 상기 가변영역 시퀀스들은 당해 기술분야에 공지된 다른 불변영역 시퀀스들에 결합된다. 중쇄 및 경쇄 가변영역들을 인코딩하는 예시적인 핵산 시퀀스들이 표 7에 제공되어 있다.
[표 6]
표 7(중쇄 및 경쇄 가변영역들을 인코딩하는 예시적인 핵산 시퀀스들)은 중쇄 및 경쇄 가변영역들을 인코딩하는 예시적인 핵산 시퀀스들을 나타내고 있으며, 여기에서 상기 가변 CDRH1, CDRH2, CDRH3, CDRLl, CDRL2 및 CDRL3 시퀀스들은 매립된다.
[표 7-1]
[표 7-2]
[표 7-3]
[표 7-4]
[표 7-5]
[표 7-6]
[표 7-7]
[표 7-8]
어떤 항원 결합 단백질들 또는 그들의 부분들(예를 들면, 전장의 항체, 중쇄 또는 경쇄, 가변도메인 또는 CDRHl, CDRH2, CDRH3, CDRLl, CDRL2 또는 CDRL3)은 cfms 또는 그들의 면역원성 단편으로 면역화된 생쥐의 B-세포들로부터 단리될 수 있다. 상기 핵산은 중합효소연쇄반응(PCR ; polymerase chain reaction) 등과 같은 통상의 절차들에 의해 단리될 수 있다. 파지 디스플레이는 공지된 기술의 다른 예이며, 이것에 의하여 항체들의 유도체들 및 다른 항원 결합 단백질들이 제조될 수 있다. 하나의 접근법에 있어서, 대상의 항원 결합 단백질의 구성요소인 폴리펩티드들이 임의의 적절한 재조합 발현 시스템 내에서 발현될 수 있으며, 발현된 폴리펩티드들은 군집되는 것이 허용되어 항원 결합 단백질 분자들을 형성한다.
표 6 및 표 7들에서 제공된 상기 핵산들은 단지 예시적인 것들이다. 유전암호(genetic code)의 퇴화도(degeneracy)로 인하여, 표 1 내지 표 4들 내에 기재된 상기 폴리펩티드 시퀀스들 또는 달리 본 명세서에서 묘사된 것들 각각은 또한 제공된 것들 이외의 다수의 다른 핵산 시퀀스들에 의해 인코딩된다. 따라서, 당해 기술분야에서 숙련된 자는 본 출원이 적절히 기술된 설명을 제공하고 그리고 각 항원 결합 단백질을 인코딩하는 각 퇴화 뉴클레오티드 시퀀스에 대한 가능성을 제공한다는 것은 이해될 수 있는 것이다.
특정의 잡종화 조건들 하에서 다른 핵산들(예를 들면, 표 6 및 표 7에 기재된 뉴클레오티드 시퀀스를 포함하는 핵산들)로 잡종화되는 핵산들을 제공하는 것이 또 다른 하나의 관점이다. 핵산들을 잡종화시키는 방법들은 당해 기술분야에서는 잘 공지된 것이다. 예를 들면, 문헌 Current Protocols in Molecular Biology, John Wiley & Sons, N.Y. (1989), 6.3.1-6.3.6을 참조하시오. 여기에서 정의된 바와 같이, 적절하게 가혹한 잡종화 조건들은 5배(5x) 염화나트륨/시트르산나트륨(SSC), 0.5% SDS, 1.0mM EDTA(pH 8.0)을 포함하는 전세척용액(prewashing solution), 약 50% 포름아미드의 잡종화 완충제(hybridization buffer), 6xSSC 및 55℃의 잡종화 온도(또는 42℃의 잡종화 온도와 함께 약 50%의 포름아미드를 포함하는 것과 같은 다른 유사한 잡종화 용액들) 및 0.5xSSC, 0.1% SDS 내에서의 60℃의 세척조건들을 사용한다. 가혹한 잡종화 조건은 45℃에서의 6xSSC 후의 68℃에서의 0.1xSSC, 0.2% SDS 내에서의 1회 또는 그 이상의 세척으로 잡종화한다. 또한, 당해 기술분야에서 숙련된 자는 서로에 대해 적어도 65%, 70%, 75%, 80%, 85%, 90%, 95%, 98% 또는 99% 동일한 뉴클레오티드 시퀀스들을 포함하는 핵산들이 전형적으로 서로 잡종화된 채로 잔류하도록 잡종화의 가혹성(stringency)이 증가되거나 또는 감소되도록 잡종화 및/또는 세척 조건들을 조작할 수 있다.
잡종화 조건들의 선택 및 적절한 조건들을 고안하기 위한 유도(guidance)에 영향을 주는 기본적인 인자들은, 예를 들면, 샘브룩, 프리트쉬 및 마니아티스(Sambrook, Fritsch, and Maniatis ; 2001, Molecular Cloning: A Laboratory Manual, Cold Spring Harbor Laboratory Press, Cold Spring Harbor, N.Y., 상기 문헌; 및 Current Protocols in Molecular Biology, 1995, 아우수벨(Ausubel)과 그의 동료들의 eds., John Wiley & Sons, Inc., sections 2.10 및 6.3-6.4)에 의해 규정되어 있으며, 예를 들어 상기 핵산의 길이 및/또는 염기 조성들에 기초하여 당해 기술분야에서 숙련된 자들에 의해 쉽게 결정될 수 있을 것이다.
핵산 내로의 돌연변이에 의해 변화들이 도입될 수 있으며, 그에 의하여 이를 인코딩하는 폴리펩티드(예를 들면, 항체 또는 항체 유도체)의 아미노산 시퀀스에서 변화되도록 유도한다. 당해 기술분야에서 공지된 임의의 기술을 이용하여 돌연변이들이 도입될 수 있다. 하나의 구체예에 있어서, 예를 들면, 사이트-지향적 돌연변이생성 프로토콜(site-directed mutagenesis protocol)을 사용하여 하나 또는 그 이상의 특정의 아미노산 잔기들이 변화된다. 다른 구체예에 있어서, 예를 들면, 무작위 돌연변이생성 프로토콜(random mutagenesis protocol)을 사용하여 하나 또는 그 이상의 무작위적으로 선택된 잔기들이 변화된다. 그러나, 이것이 만들어지면, 돌연변이 폴리펩티드는 원하는 특성에 대하여 발현되고 그리고 스크리닝될 수 있다.
인코딩하는 폴리펩티드의 생물학적 활성을 명백하게 변화시킴이 없이 핵산 내로 돌연변이들을 도입할 수 있다. 예를 들면, 비-필수 아미노산 잔기들에서 아미노산 치환되도록 유도하는 뉴클레오티드 치환을 할 수 있다. 달리, 하나 또는 그 이상의 돌연변이들이 인코딩하는 폴리펩티드의 생물학적 활성을 선택적으로 변화시키는 핵산 내로 도입될 수 있다. 예를 들면, 상기 돌연변이는 상기 생물학적 활성을 정량적으로 또는 정성적으로 변화시킬 수 있다. 정량적인 변화들의 예들에는 상기 활성의 증가, 감소 또는 제거들이 포함된다. 정성적인 변화들의 예들에는 항체의 항원 특이성의 변화가 포함된다. 하나의 구체예에 있어서, 본 명세서에서 기술된 임의의 항원 결합 단백질을 인코딩하는 핵산이 돌연변이되어 당해 기술분야에서 잘 구축된 분자생물학 기술들을 사용하여 상기 아미노산 시퀀스를 변형시킬 수 있다. 예를 들면, 실시예 4는 어떻게 핵산 시퀀스들(표 6을 참조)이 하나 또는 그 이상의 아미노산 치환들을 특정의 항원 결합 단백질들 내로 도입시켜 항원 결합 단백질들 1.2 SM 1.109 SM 및 2.360 SM들을 생산하도록 돌연변이 되는 지를 기술하고 있다. 다른 돌연변이들을 포함하는 부가의 항원 결합 단백질들이 유사한 방법으로 생산될 수 있다.
핵산 시퀀스들의 검출을 위한 프라이머 또는 잡종화 탐침들로서 사용되기에 적절한 핵산 분자들을 제공하는 것이 본 발명의 또 다른 관점이다. 핵산 분자는 단지 전장의 폴리펩티드, 예를 들면, 폴리펩티드의 활성부분(예를 들면, c-fms 결합 부분)을 인코딩하는 탐침 또는 프라이머 또는 단편으로 사용될 수 있는 단편을 인코딩하는 핵산의 부분을 포함할 수 있다.
핵산의 시퀀스를 기초로 하는 탐침들은 상기 핵산 또는 유사한 핵산들, 예를 들면, 폴리펩티드를 인코딩하는 전사체들을 검출하는 데 사용될 수 있다. 상기 탐침은 표지기, 예를 들면, 방사성동위원소, 형광 화합물, 효소 또는 효소 보조인자를 포함할 수 있다. 이러한 탐침들은 상기 폴리펩티드를 발현하는 세포를 동정하는 데 사용될 수 있다.
폴리펩티드 또는 그들의 부분(예를 들면, 하나 또는 그 이상의 CDRs들 또는 하나 또는 그 이상의 가변영역 도메인들)을 인코딩하는 핵산을 포함하는 벡터들을 제공하는 것이 본 발명의 또 다른 관점이다. 벡터들의 예들에는 플라스미드, 바이러스벡터(viral vectors), 비-에피좀의 포유동물 벡터들 및 발현 벡터들, 예를 들면, 재조합 발현 벡터들이 포함되나, 이들에 제한되는 것은 아니다. 상기 재조합 발현 벡터들은 숙주세포 내의 핵산의 발현에 적절한 형태의 핵산을 포함할 수 있다. 상기 재조합 발현 벡터들은 발현을 위하여 사용될 상기 숙주세포들을 기반으로 선택되는 하나 또는 그 이상의 조절 시퀀스들을 포함하며, 이는 발현될 상기 핵산 시퀀스에 작동가능하게 연결된다. 조절 시퀀스들에는 여러 형태들의 숙주세포들 내에서의 뉴클레오티드 시퀀스의 직접적 구성 발현을 하는 것들(예를 들면, SV49 초기 유전자 인핸서, 로우스 육종 바이러스 프로모터(Rous sarcoma virus promoter) 및 거대세포바이러스 프로모터(cytomegalovirus promoter)), 단지 특정의 숙주세포들 내에서의 상기 뉴클레오티드 시퀀스의 직접적 발현을 하는 것들(예를 들면, 조직-특이성의 조절 시퀀스들, 본 명세서에서 그들 전체들을 참조로 인용하는 보스(Voss)와 그의 동료들의 1986, Trends Biochem. Sci. 11:287, 마니아티스(Maniatis)와 그의 동료들의 1987, Science 236:1237들을 참조) 및 특정의 처리 또는 조건에 대응하는 뉴클레오티드 시퀀스의 직접적 유도가능 발현을 하는 것들(예를 들면, 포유동물 세포들 내에서의 메탈로티오닌 프로모터(metallothionin promoter) 및 원핵계 및 진핵계 둘 다에서의 티이티-반응성(tet-responsive) 및/또는 스트렙토마이신 반응성 프로모터(동일 문헌 참조)들이 포함된다. 당해 기술분야에서 숙련된 자들에게는 상기 발현 벡터의 설계가 형질전환되어야 할 숙주세포의 선택, 원하는 단백질의 발현의 수준 등의 선택에 따라 이러한 인자들에 의존적일 수 있다는 것은 이해될 수 있는 것이다. 상기 발현 벡터들은 숙주세포들 내로 도입될 수 있으며, 그에 의하여 본 명세서에서 기술된 바와 같은 핵산들에 의해 인코딩된 융합 단백질 또는 펩티드들을 포함하는 단백질들 또는 펩티드들을 생산할 수 있다.
재조합 발현 벡터가 그 안으로 도입된 숙주세포들을 제공하는 것이 본 발명의 또 다른 관점이다. 숙주세포는 임의의 원핵세포(예를 들면, 대장균) 또는 진핵세포(예를 들면, 효모, 곤충 또는 포유동물 세포들(예를 들면, CHO 세포들))이 될 수 있다. 벡터 DNA는 통상의 형질전환 또는 형질변환 기술들을 통하여 원핵세포 또는 진핵세포들 내로 도입될 수 있다. 포유동물 세포들의 안정한 형질변환을 위하여, 발현 벡터 및 사용되는 형질변환 기술에 따라, 세포들의 단지 작은 부분(fraction)이 그들의 게놈 내로 상기 외래 DNA와 통합(integrate)될 수 있다. 이들 통일체 구성부분(integrants)들을 동정하고 그리고 선택하기 위하여, 선택가능한 마커(marker)를 인코딩하는 유전자(예를 들면, 항생제에 대한 저항을 위한)가 대체로 대상의 상기 유전자와 함께 상기 숙주세포들 내로 도입된다. 바람직한 선택가능한 마커들에는 G418, 하이그로마이신(hygromycin) 및 메토트렉세이트(methotrexate) 등과 같이 약물에 대한 저항성을 부여하는 것들이 포함된다. 상기 도입된 핵산들로 안정하게 형질변환된 세포들은 다른 방법들 중에서도 약물선택(drug selection ; 예를 들면, 상기 선택가능한 마커 유전자가 내포된 세포들은 생존할 수 있는 반면에 다른 세포들은 사멸한다)에 의해 동정될 수 있다.
항원 결합 단백질들의 제조
제공될 수 있는 비-인간의 항체들은, 예를 들면, 생쥐, 집쥐, 토끼, 염소, 당나귀 또는 비-인간의 영장류(예를 들면, 사이노몰거스 또는 레서스 원숭이(rhesus monkey) 또는 유인원(예를 들면, 침팬지) 등과 같은 임의의 항체-생성 동물들로부터 유도될 수 있다. 비-인간의 항체들은, 예를 들면, 시험관 내 세포 배양 및 세포-배양 기반 응용예들, 또는 상기 항체에 대한 면역반응이 일어나지 않거나 또는 불분명하거나, 방지되거나, 문제되지 않거나 또는 소망되는 임의의 다른 응용예에 사용될 수 있다. 특정의 구체예들에 있어서, 상기 항체들은 전장의 c-fms 또는 c-fms의 상기 세포외 도메인으로 면역화시키는 것에 의하여 생산될 수 있다. 달리, 특정의 비-인간의 항체들은 본 명세서에서 제공되는 특정의 항체들이 결합하는 상기 에피토프의 일부를 형성하는 c-fms의 단편들인 아미노산들로 면역화시키는 것에 의해 야기될 수 있다(위의 문헌 참조) 상기 항체들은 폴리클로날, 모노클로날이 될 수 있거나 또는 재조합 DNA를 발현시키는 것에 의해 숙주세포들 내에서 합성될 수 있다.
완전한 인간 항체들은 인간 면역글로블린 로커스들을 포함하는 이식유전자를 가진 동물들을 면역화시키는 것에 의하여 또는 인간 항체들의 전목록을 발현하는 파지 디스플레이 라이브러리를 선택하는 것에 의하여 앞서 기술한 바와 같이 제조될 수 있다.
상기 모노클로날 항체들(mAbs)은 통상의 모노클로날 항체 방법론, 예를 들면, 콜러와 밀스타인(Kohler and Milstein)의 1975, Nature 256:495의 표준 체세포 잡종화 기술을 포함하는 다양한 기술들에 의해 생산될 수 있다. 달리, 모노클로날 항체들을 생산하는 다른 기술들, 예를 들면, B-림프구들의 바이러스 또는 종양유발 형질전환이 사용될 수 있다. 하이브리도마들을 제조하기 위한 하나의 적절한 동물계는 쥣과이며, 이는 매우 잘 구축된 절차이다. 융합을 위한 면역화된 비장 세포들의 단리를 위한 면역화 프로토콜 및 기술들은 당해 기술분야에서 공지이다. 이러한 절차들을 위하여는, 면역화된 생쥐로부터의 B세포들이 쥣과의 동물의 골수종 세포주 등과 같은 적절한 불멸화된 융합짝(fusion partner)와 함께 융합된다. 원하는 경우, 생쥐 대신에 집쥐들 또는 이외의 다른 포유동물들이 면역화될 수 있으며, 이러한 동물들로부터의 B세포들이 상기 쥣과의 동물의 골수종 세포주와 함께 융합되어 하이브리도마들을 형성할 수 있다. 달리, 쥐와는 다른 근원으로부터의 골수종 세포주가 사용될 수 있다. 하이브리도마들을 만들기 위한 융합 절차들 역시 잘 알려져 있다.
제공되는 상기 단일쇄 항체들은 아미노산 브릿지(짧은 펩티드 링커)를 통하여 중쇄 및 경쇄 가변 도메인(Fv 영역) 단편들을 연결시키는 것에 의해 형성되어 단일의 폴리펩티드쇄의 결과를 가져온다. 이러한 단일-쇄 Fvs(scFvs)는 상기 두 가변 도메인 폴리펩티드(VL 및 VH)들을 인코딩하는 DNA들 사이의 펩티드 링커를 인코딩하는 DNA를 융합시키는 것에 의해 제조될 수 있다. 그 결과의 폴리펩티드들은 그 자체 상으로 다시 중첩되어 항원-결합 단량체들을 형성하거나 또는 이들은 상기 두 가변 도메인들 사이의 가요성의 링커의 길이에 따라 복합체(multimers ; 예를 들면, 이량체, 삼량체 또는 사량체들)들로 형성될 수 있다(코르트(Kortt)와 그의 동료들의 1997, Prot. Eng. 10:423; 코르트와 그의 동료들의 2001, Biomol. Eng. 18:95-108). 서로 다른 VL 및 VH-함유 폴리펩티드들을 결합하는 것에 의하여, 서로 다른 에피토프들에 결합하는 복합체성 scFvs를 형성할 수 있다(크리앙쿰(Kriangkum)과 그의 동료들의 2001, Biomol. Eng. 18:31-40). 단일쇄 항체들의 생산을 위하여 개발된 기술들에는 미합중국 특허 제4,946,778호; 버드의 1988, Science 242:423; 허스톤과 그의 동료들의 1988, Proc. Natl. Acad. Sci. U.S.A. 85:5879; 와드(Ward)와 그의 동료들의 1989, Nature 334:544, 드 그라아프(de Graaf)와 그의 동료들의 2002, Methods Mol Biol. 178:379-387들에 기술된 것들이 포함된다. 본 명세서에서 제공되는 항체들로부터 유도되는 단일쇄 항체들에는 표 2에 묘사된 중쇄 및 경쇄 가변영역들의 가변 도메인 조합들을 포함하는 scFvs 또는 표 3 및 표 4에 묘사된 CDRs들을 포함하는 경쇄 및 중쇄 가변 도메인들의 조합들이 포함되나, 이들에 제한되는 것은 아니다.
본 명세서에서 제공되는 하나의 서브클래스인 항체들은 서브클래스 스위칭법(subclass switching methods)들을 사용하여 다른 서브클래스로부터의 항체들로 변화될 수 있다. 따라서, IgG 항체들은, 예를 들면, IgM 항체로부터 유도될 수 있으며, 그 역 또한 가능하다. 이러한 기술들은 주어진 항체(부모 항체)의 항원 결합 특성들을 보유하는 새로운 항체들의 제조를 가능하게 하나, 또한 상기 부모 항체와는 다른 항체 아이소타입 또는 서브클래스와 연관된 생물학적 특성들을 나타낸다. 재조합 DNA 기술들이 사용될 수 있다. 특정의 항체 폴리펩티드들을 인코딩하는 복제된 DNA, 예를 들면, 원하는 아이소타입의 항체의 상기 불변 도메인을 인코딩하는 DNA가 이러한 절차들에서 사용될 수 있다. 예를 들면, 란토(Lantto)와 그의 동료들의 2002, Methods Mol. Biol. 178:303-316을 참조하시오.
따라서, 제공되는 상기 항체들은, 예를 들면, 원하는 아이소타입(예를 들면, IgA, IgGl, IgG2, IgG3, IgG4, IgE 및 IgD) 및 마찬가지로 이들의 Fab 또는 F(ab')2 단편들을 갖는 앞서 기술한 상기 가변 도메인 조합들을 포함하는 것들이 포함된다. 더욱이, IgG4가 희망되는 경우, 여기에 참조로 인용되는 블룸(Bloom)과 그의 동료들의 1997, Protein Science 6:407에서 기술된 바와 같은 상기 힌지 영역 내에 점 돌연변이를 도입하여 상기 IgG4 항체들 내의 이종성(heterogeneity)을 야기할 수 있는 내부-H 쇄 이황화 결합(intra-H chain disulfide bonds)들을 형성하려는 경향을 완화시키는 것이 또한 희망될 수 있다.
더욱이, 서로 다른 특성들(예를 들면, 이들이 결합하는 상기 항원에 대한 친화도를 변화시키는 것)을 갖는 항체들을 유도하기 위한 기술들이 또한 공지되어 있다. 체인셔플링(chain shuffling)으로 언급되는 이러한 하나의 기술은 종종 파지 디스플레이로 언급되는 심질의 박테리오파지의 표면에 대한 면역글로블린 가변 도메인 유전자 전목록을 디스플레이하는 것을 포함한다. 체인셔플링은 마르크스(Marks)와 그의 동료들의 1992, BioTechnology 10:779에 의해 기술된 바와 같이 합텐 2-페닐옥사졸-5-온에 대한 높은 친화도 항체들을 준비하는 데 사용되었다.
표 2에 기술된 상기 중쇄 및 경쇄 가변영역들 또는 표 3 및 표 4에 기술된 상기 CDRs들(및 상기 인코딩 핵산에 대하여 대응하는 변형들)에 대하여 보존적 변형들이 이루어져서 기능적이고 그리고 생화학적 특성들을 갖는 c-fms 항원 결합 단백질을 생산할 수 있다. 이러한 변형들을 달성하기 위한 방법들은 앞서 기술되었다.
C-fms 항원 결합 단백질들은 여러 방법들로 더 변형될 수 있다. 예를 들면, 이들이 치료적인 목적들로 사용되어야 하는 경우, 이들은 폴리에틸렌글리콜과 콘쥬게이트되어(피이지화) 혈청 반감기를 연장하거나 또는 단백질 전달을 증강시킬 수 있다. 달리, 상기 대상체 항체들 또는 그들의 단편들의 상기 V영역은 다른 항체 분자의 상기 Fc 영역에 융합될 수 있다. 이러한 목적을 위하여 사용되는 상기 Fc 영역은 변형되어 이것이 보체와 결합하지 않도록 할 수 있으며, 따라서 상기 융합 단백질이 치료약물로서 사용되는 경우 환자 내에서 세포 용해 유발의 가능성을 감소시키도록 할 수 있다. 게다가, 상기 대상체 항체들 또는 그들의 기능적인 단편들은 인간 혈청 알부민과 콘쥬게이트되어 상기 항체 또는 그들의 단편의 혈청 반감기를 증강시킬 수 있다. 상기 항원 결합 단백질들 또는 이들의 단편들에 대한 다른 유용한 융합짝은 티록신결합전알부민(TTR)이다. 티록신결합전알부민은 사량체를 형성하는 능력을 가지며, 따라서 항체-TTR 융합 단백질은 그의 결합 활성(binding avidity)를 증가시킬 수 있는 다가(multivalent)의 항체를 형성할 수 있다.
달리, 본 명세서에서 기술되는 상기 항원 결합 단백질들의 기능적 및/또는 생화학적 특성들에서의 실제적인 변형들은 (a) 치환의 영역 내에서의 분자 골격의 구조, 예를 들면, 시트 또는 헬리컬 형태, (b) 목표 사이트에서의 상기 분자의 하전 또는 소수성 또는 (c) 측쇄의 부피 크기(bulkiness)를 유지하는 데 대한 그들의 영향에서의 명백하게 다른 상기 중쇄 및 경쇄들의 상기 아미노산 시퀀스 내에서의 치환을 생성하는 것에 의해 달성될 수 있다. "보존적 아미노산 치환"은 자연의 아미노산 잔기의 그 위치에서의 상기 아미노산 잔기의 극성 또는 하전에 대하여 거의 또는 전혀 영향이 없는 비자연적인 잔기로의 치환을 포함한다. 위의 표 3을 참조하시오. 더욱이, 상기 폴리펩티드 내의 임의의 자연의 잔기는 또한 알라닌 스캐닝 돌연변이생성에 대해 이미 기술한 바와 같이 알라닌으로 치환될 수 있다.
대상 항체들의 아미노산 치환들(보존적 또는 비-보존적인 지를 불문)은 통상적인 기술들을 적용시키는 것에 의해 당해 기술분야에서 숙련된 자들에 의해서 실행될 수 있다. 아미노산 치환들은 본 명세서에서 제공되는 상기 항체들의 중요한 잔기들을 동정하거나 또는 인간 c-fms에 대한 이들 항체들의 친화도를 증가시키거나 감소시키거나 또는 본 명세서에서 기술되는 다른 항원-결합 단백질들의 결합 친화도를 변화시키기 위하여 사용될 수 있다.
항원 결합 단백질들을 발현시키는 방법들
본 명세서에서 기술되는 바와 같은 적어도 하나의 폴리뉴클레오티드를 포함하는 발현 시스템들 및 플라스미드, 발현 벡터, 전사 또는 발현 카셋트(transcription or expression cassettes)들의 형태의 구조물들이 또한 본 명세서에서 제공되며, 이러한 발현 시스템들 또는 구조물들을 포함하는 숙주세포들도 마찬가지이다.
본 명세서에서 제공되는 상기 항원 결합 단백질들은 다수의 통상적인 기술들 중의 임의의 것에 의하여 제조될 수 있다. 예를 들면, c-fms 항원 결합 단백질들은 당해 기술분야에서 공지된 임의의 기술을 사용하여 재조합 발현 시스템들에 의해 생산될 수 있다. 예를 들면, 모노클로날 항체들, 하이브리도마들(Monoclonal Antibodies, Hybridomas : A New Dimension in Biological Analyses, 케네트(Kennet)와 그의 동료들(저) Plenum Press, New York (1980); 및 항체들( Antibodies : A Laboratory Manual, 할로우와 레인(Harlow and Lane)(저), Cold Spring Harbor Laboratory Press, Cold Spring Harbor, N. Y. (1988)들을 참조하시오.
항원 결합 단백질들은 하이브리도마 세포주들(예를 들면, 특히 항체들은 하이브리도마들 내에서 발현될 수 있다) 내에서 또는 하이브리도마들 이외의 세포주들 내에서 발현될 수 있다. 상기 항체들을 인코딩하는 발현 구조물들이 포유동물, 곤충 또는 미생물 숙주세포를 변형시키는 데 사용될 수 있다. 형질전환은, 예를 들면, 미합중국 특허 제4,399,216호; 동 제4,912,040호; 동 제4,740,461호; 동 제4,959,455호에 의해 예시된 바와 같이, 폴리뉴클레오티드를 바이러스 또는 박테리오파지 내에 패키징(packaging) 하고 그리고 당해 기술분야에서 공지된 형질변환 절차들에 의해 숙주세포를 구조물로 형질도입하는 것을 포함하여 폴리뉴클레오티드들을 숙주세포 내로 도입하기 위한 임의의 공지된 방법을 사용하여 수행될 수 있다. 사용되는 최적의 형질전환 절차들은 어느 형태의 숙주세포가 형질전환되는 지에 의존적일 수 있다. 이종기원의 폴리펩티드들을 포유동물 세포들 내로 도입하기 위한 방법들은 당해 기술분야에서 공지되어 있으며, 덱스트란-매개 형질변환(dextran-mediated transfection), 칼슘포스페이트 침강(calcium phosphate precipitation), 폴리브렌 매개 형질변환(polybrene mediated transfection), 원형질체 융합(protoplast fusion), 전기천공(electroporation), 리포좀들 내로의 폴리뉴클레오티드(들)의 캡슐화(encapsulation of the ρolynucleotide(s) in liposomes), 핵산과 양성적으로-하전된 지질들과의 혼합(mixing nucleic acid with positively-charged lipids) 및 상기 DNAd의 핵종 내로의 직접적 미세주사(direct microinjection of the DNA into nuclei)들이 포함되나, 이들에 제한되는 것은 아니다.
재조합 발현 구조물들은 전형적으로 본 명세서에서 제공되는 하나 또는 그 이상의 CDRs들; 경쇄 불변영역; 경쇄 가변영역; 중쇄 불변영역(예를 들면, CH1, CH2 및/또는 CH3); 및/또는 c-fms 항원 결합 단백질의 다른 스캐폴드 부(scaffold portion)들 중의 하나 또는 그 이상을 포함하는 폴리펩티드를 인코딩하는 핵산 분자를 포함한다. 이들 핵산 시퀀스들은 표준 결착 기술(standard ligation techniques)들을 사용하여 적절한 발현 벡터 내로 삽입된다. 하나의 구체예에 있어서, 상기 중쇄 또는 경쇄 불변영역은 상기 항-c-fms-특이적 중쇄 또는 경쇄 가변영역의 상기 C-말단에 부착되고 그리고 발현 벡터 내로 결착된다. 상기 벡터는 전형적으로 사용되는 특정의 숙주세포 내에서 기능적인 것으로 선택된다(즉, 상기 벡터는 상기 숙주세포 기구(host cell machinary)와 경쟁가능하며 유전자의 증폭 및/또는 발현이 일어날 수 있도록 하는 것을 허용한다). 일부 구체예들에 있어서, 디히드로폴레이트 리덕타아제(dihydrofolate reductase) 등과 같은 단백질 리포터(protein reporters)들을 사용하는 단백질-단편 상보성 분석법(protein-fragment complementation assays)들을 사용하는 벡터들이 사용된다(예를 들면, 본 명세서에서 참조로 인용하는 미합중국 특허 제6,270,964호를 참조하시오). 적절한 발현 벡터들은, 예를 들면, 인비트로젠 라이프 테크놀로지스(Invitrogen Life Technologies) 또는 비디 바이오사이언시즈(BD Biosciences)(이전의 "클론테크(Clontech)")들로부터 구입될 수 있다. 상기 항체들 및 단편들의 복제 및 발현을 위한 다른 유용한 벡터들에는 비앙키와 맥그류(Bianchi and McGrew)의 2003, Biotech. Biotechnol. Bioeng. 84:439-44에서 기술된 것들이 포함되며, 이를 본 명세서에서 참조로 인용한다. 별도의 적절한 발현 벡터들이, 예를 들면, 문헌 Methods Enzymol., vol. 185 (D. V. Goeddel, ed.), 1990, New York: Academic Press에서 논의되어 있다.
전형적으로, 상기 임의의 숙주세포들에서 사용되는 발현 벡터들은 플라스미드 유지 및 외인성의 뉴클레오티드 시퀀스들의 복제 및 발현을 위한 시퀀스들을 포함할 수 있다. 특정의 구체예들에서 집단적으로 "측면서열(flanking sequences)"로 언급되는 이러한 시퀀스들은 전형적으로는 프로모터, 하나 또는 그 이상의 인핸서 시퀀스들, 복제의 원본(origin), 전사적종결시퀀스(transcriptional termination sequence), 도너 및 억셉터 스플라이스 사이트를 포함하는 완전한 인트론 시퀀스(complete intron sequence containing a donor and acceptor splice site), 폴리펩티드 분비를 위한 리더 시퀀스를 인코딩하는 시퀀스, 리보좀 결합 사이트, 폴리아데닐화 시퀀스(polyadenylation sequence), 발현될 폴리펩티드를 인코딩하는 핵산을 삽입하기 위한 폴리링커 영역 및 선택가능한 마커 요소(marker element)들 중의 하나 또는 그 이상을 포함할 수 있다. 이들 시퀀스들 각각을 이하에서 기술한다.
선택적으로, 상기 벡터는 "태그"-인코딩 시퀀스, 즉, 시퀀스를 코딩하는 상기 c-fms 항원 결합 단백질의 5' 또는 3' 단부에 위치하는 올리고뉴클레오티드 분자를 포함할 수 있으며, 상기 올리고뉴클레오티드 시퀀스는 polyHis(hexaHis 등과 같은) 또는 FLAG®, HA(헤마토글루티닌 인플루엔자 바이러스) 또는 myc들을 인코딩하며, 이들에 대한 상용적으로 획득가능한 항체들이 존재한다. 이 태그는 전형적으로 상기 폴리펩티드의 발현에 의하여 상기 폴리펩티드에 융합되며, 상기 숙주세포로부터의 상기 c-fms 항원 결합 단백질의 친화도 정제 또는 검출을 위한 수단으로서 기능할 수 있다. 친화도 정제는, 예를 들면, 친화도 매트릭스(affinity matrix)로서 상기 태그에 대한 항체들을 사용하는 컬럼크로마토그래피에 의해 수행될 수 있다. 선택적으로, 상기 태그는 개열을 위한 특정의 펩티다아제들을 사용하는 것과 같은 여러 수단에 의해 후속적으로 상기 정제된 c-fms 항원 결합 단백질로부터 제거될 수 있다.
측면서열이 상기 숙주세포 기구 내에서 기능적이고 그리고 상기 기구에 의해 활성화될 수 있는 한, 상기 측면서열들은 동종기원(즉, 상기 숙주세포와 동일한 종들 및/또는 스트레인), 이종기원(즉, 상기 숙주세포 종들 또는 스트레인과는 다른 종들로부터의), 하이브리드(즉, 하나 이상의 원천으로부터의 측면서열들의 조합), 합성 또는 천연의 것이 될 수 있다. 마찬가지로, 측면서열이 상기 숙주세포 기구 내에서 기능적이고 그리고 상기 기구에 의해 활성화될 수 있는 한, 상기 측면서열의 원천은 임의의 원핵 또는 진핵 유기체, 임의의 척추 유기체 또는 무척추 유기체, 또는 임의의 식물들이 될 수 있다.
상기 벡터들 내에서 유용한 측면서열들은 당해 기술분야에서 공지된 여러 방법들 중 임의의 것에 의해 수득될 수 있다. 전형적으로, 본 명세서에서 유용한 측면서열들은 매핑(mapping)에 의해 및/또는 제한효소 소화(restriction endonuclease digestion)에 의해 앞서 동정될 수 있으며, 따라서 적절한 제한효소들을 사용하여 적절한 조직 원천으로부터 단리될 수 있다. 일부 경우들에 있어서, 측면서열의 완전한 뉴클레오티드 시퀀스는 공지된 것일 수 있다. 여기에서, 상기 측면서열은 핵산 합성 또는 복제에 대하여 본 명세서에서 기술된 방법들을 사용하여 합성할 수 있다.
상기 측면서열의 전부 또는 일부가 공지되었는 지에 무관하게, 중합효소연쇄반응(PCR)을 사용하여 및/또는 동일 또는 다른 종들로부터의 올리고뉴클레오티드 및/또는 측면서열 단편 등과 같은 적절한 탐침으로 게놈 라이브러리를 스크리닝하는 것에 의해 수득될 수 있다. 상기 측면서열이 공지되지 않은 경우, 측면서열을 포함하는 DNA의 단편이 예를 들면 코딩 시퀀스 또는 심지어 다른 유전자나 유전자들을 포함할 수 있는 DNA의 보다 큰 조각으로부터 단리될 수 있다. 단리는 적절한 DNA 단편을 생성하기 위한 제한효소 소화와 후속하는 아가로스 겔(agarose gel) 정제인 키아겐 컬럼 크로마토그래피(Qiagen column chromatography)(미합중국 캘리포니아주 채스워스(Chatsworth) 소재) 또는 숙련된 기술자에 공지된 다른 방법들을 사용하는 단리에 의하여 수행될 수 있다. 이러한 목적을 수행하기 위한 적절한 효소들의 선택은 당해 기술분야에서 숙련된 자들에게는 쉽게 이해될 수 있을 것이다.
복제의 원본은 전형적으로 상용저긍로 구입하는 이들 원핵 발현 벡터들의 일부이고, 그리고 상기 원본은 숙주세포 내에서의 상기 벡터의 증폭을 돕는다. 상기 선택된 벡터가 복제 사이트의 원본을 포함하지 않는 경우, 공지된 시퀀스를 기초하여 화학적으로 합성하고 그리고 상기 벡터 내로 결착시킬 수 있다. 예를 들면, 복제의 원본은 플라스미드 pBR322(미합중국 매사추세츠 베벌리 소재 뉴잉글랜드 바이오랩스(New England Biolabs))가 대부분의 그램-음성의 박테리아에 대하여 적절하며, 여러 바이러스 원본들(예를 들면, SV40, 폴리오마(polyoma), 아데노바이러스, 수포성 구내염 바이러스(VSV ; vesicular stomatitus virus) 또는 HPV 또는 BPV 등과 같은 파필로마바이러스(papillomaviruses))이 포유동물 세포들 내에서 벡터들을 복제하는 데 유용하다. 대체로, 복제 구성요소의 원본은 포유동물 발현 벡터들(예를 들면, 상기 SV40 원본은 이것이 또한 상기 바이러스 초기 프로모터를 포함하기 때문에 종종 사용되고 있다)에 대하여는 필요치 않다.
전사 종결 시퀀스는 전형적으로 폴리펩티드 코딩 영역의 3'에서 단부까지에 위치되며, 전사를 종결시키는 것으로 기능한다. 대개, 원핵세포들 내의 전사 종결 시퀀스는 G-C 풍부 단편이며 폴리-T 시퀀스가 후속한다. 비록 상기 시퀀스가 라이브러리로부터 쉽게 복제되거나 또는 심지어 벡터의 일부로서 상용적으로 구입될 수 있음에도 불구하고, 이는 또한 본 명세서에서 기술된 바와 같은 핵산 합성을 위한 방법을 사용하여 쉽게 합성된다.
선택가능한 마커 유전자는 선택적인 배지 내에서 성장한 숙주세포의 생존 및 성장을 위하여 필요한 단백질을 인코딩한다. 전형적인 선택 마커 유전자들은 (a) 다른 항생제들 또는 독소들, 예를 들면, 원핵 숙주세포들에 대한 암피실린, 테트라사이클린 또는 가나마이신들에 저항성을 부여하거나; (b) 상기 세포의 영양요구 결핍(auxotrophic deficiencies)을 보충하거나; 또는 (c) 복합체 또는 정의된 매질로부터는 획득할 수 없는 절대적인 영양소(critical nutrients)들을 공급하는 단백질들을 인코딩한다. 특정의 선택가능한 마커들은 가나마이신 저항성 유전자, 암피실린 저항성 유전자 및 테트라사이클린 저항성 유전자들이다. 유리하게도, 네오마이신 저항성 유전자가 또한 원핵 및 진핵 숙주세포들 둘 다에서의 선택에 대하여 사용될 수 있다.
다른 선택가능한 유전자들이 발현될 유전자를 증폭하는 데 사용될 수 있다. 증폭은 성장 또는 세포 생존에 절대적인 단백질의 생산을 위하여 요구되는 유전자들이 재조합 세포들의 연속하는 세대들의 크로모좀들 내에서 이중으로 반복되는 과정이다. 포유동물 세포들에 대한 적절한 선택가능한 마커들의 예들에는 디히드로폴레이트 리덕타아제(dihydrofolate reductase ; DHFR) 및 무프로모터 티미딘 키나아제 유전자(promoterless thymidine kinase genes)들이 포함된다. 포유동물 세포 형질전환체(transformants)들은 선택압 하에 놓여지며, 여기에서 단지 형질전환체들은 독특하게 상기 벡터 내에 존재하는 상기 선택가능한 유전자의 덕택으로 생존하도록 적응된다. 선택압은 상기 형질전환된 세포들을 상기 매질 내의 선택시약(selection agent)의 농도가 충분하게 증가되고 그에 의하여 상기 선택가능한 유전자 및 c-fms 폴리펩티드에 결합하는 항원 결합 단백질 등과 같은 다른 유전자를 인코딩하는 상기 DNA들 둘 다의 증폭을 야기하는 조건들 하에서 배양시키는 것에 의하여 부여된다. 그 결과, 항원 결합 단백질 등과 같은 폴리펩티드의 증가된 양들이 상기 증폭된 DNA로부터 합성되게 된다.
리보좀-결합 사이트는 대개 mRNA의 전사 개시를 위하여 필요하며, 샤인-달가노 시퀀스(Shine-Dalgarno sequence)(원핵세포들) 또는 코작 시퀀스(Kozak sequence)(진핵세포)에 의해 특정화된다. 상기 요소는 전형적으로 발현되어야 할 상기 폴리펩티드의 3'에서 상기 프로모터까지 그리고 5'에서 상기 코딩 시퀀스까지에 위치된다.
진핵의 숙주세포 발현 시스템 내에서 글리코실화가 소망되는 경우와 같은 일부 경우들에 있어서, 글리코실화 또는 수율을 증가시키기 위하여 여러 프리-시퀀스 또는 프로-시퀀스(pre- or pro-sequences)들을 조작할 수 있다. 예를 들면, 특정의 시그널 펩티드의 펩티다아제 개열 사이트를 바꾸거나 또는 프로시퀀스들을 첨가할 수 있으며, 이들은 또한 글리코실화에 영향을 줄 수 있다. 최종 단백질 생성물은 -1 위치(성숙한 단백질의 첫 번째의 아미노산에 대하여)에 발현에 관여하는 하나 또는 그 이상의 부가의 아미노산들을 가질 수 있으며, 이는 완전히 제거되지는 않을 것이다. 예를 들면, 최종 단백질 생성물은 상기 아미노-말단에 부착된, 상기 펩티다아제 개열 사이트에서 발견되는 하나 또는 두 개의 아미노산 잔기들을 가질 수 있다. 달리, 일부 효소 개열 사이트들의 사용은, 상기 효소가 상기 성숙한 폴리펩티드 내에서의 이러한 영역에서 절단하는 경우, 원하는 폴리펩티드의 약간 끝이 잘려진 형태의 결과를 가져올 수 있다.
발현 및 복제는 전형적으로 상기 숙주 유기체에 의해 인식되고 그리고 c-fms 항원 결합 단백질을 인코딩하는 분자에 작동가능하게 연결되는 프로모터를 포함할 것이다. 프로모터들은 구조적 유전자의 전사를 제어하는 상기 구조적 유전자(대체로 대략 100 내지 1,000bp)의 개시 코돈에 대하여 업스트림(즉, 5')에 위치하는 전사되지 않는 시퀀스들이다. 프로모터들은 통상적으로 유도성 프로모터(inducible promoters) 및 구성성 프로모터(constitutive promoters)의 두 클래스들로 그룹지어진다. 유도성 프로모터들은 영양소의 존재 또는 부재 또는 온도에서의 변화 등과 같은 배양 조건들에서의 일부 변화에 대응하는 그드르ㅣ 조절 하에서의 DNA로부터의 증가된 수준들의 전사를 개시한다. 반면에, 구성성 프로모터들은 일정하게 즉, 유전자 발현에 대하여 조절이 거의 또는 전혀 없이 그들이 작동가능하게 연결되는 유전자를 전사한다. 다양한 잠재적인 숙주세포들에 의하여 인식되는 다수의 프로모터들이 잘 알려져 있다. 적절한 프로모터는 제한효소 소화 및 벡터 내로 원하는 프로모터 시퀀스를 삽입하는 것에 의하여 원천 DNA로부터 상기 프로모터를 제거하는 것에 의하여 c-fms 항원 결합 단백질을 포함하는 중쇄 또는 경쇄를 인코딩하는 DNA에 작동가능하게 연결된다.
효모 숙주들에 대하여 사용하기에 적절한 프로모터들이 또한 당해 기술분야에서 잘 알려져 있다. 효모 인핸서들이 요모 프로모터들과 함께 유리하게 사용된다. 포유동물 숙주세포들에 대하여 사용되기 위한 적절한 프로모터들은 잘 알려져 있으며, 폴리오마 바이러스, 계두 바이러스(fowlpox virus), 아데노바이러스(아데노바이러스 2 등과 같은), 우파필로마바이러스(bovine papilloma virus), 조류육종바이러스(avian sarcoma virus), 거대세포바이러스, 레트로바이러스. 비형 간염 바이러스 및 시미안 바이러스 40(Simian Virus 40 ; SV40) 등과 같은 바이러스들의 유전자들로부터 수득되는 것들이 포함되나, 이들에 제한되는 것은 아니다. 다른 적절한 포유동물 프로모터들에는 이종기원의 포유동물 프로모터들, 예를 들면, 열-충격 프로모터(heat-shock promoters)들 및 액틴 프로모터(actin promoter)들이 포함된다.
흥미의 대상이 될 수 있는 별도의 프로모터들에는 SV40 초기 프로모터(베노이스트와 챔본(Benoist and Chambon)의 1981, Nature 290:304-310); CMV 프로모터(쏘른센(Thornsen)과 그의 동료들의 1984, Proc. Natl. Acad. U.S.A. 81:659-663); 로우스 육종 바이러스의 3' 장말단 반복 내에 포함되는 프로모터(야마모토(Yamamoto)와 그의 동료들의 1980, Cell 22:787-797); 헤르페스 티미딘 키나아제 프로모터(와그너와 그의 동료들의 1981, Proc. Natl. Acad. Sci. U.S.A. 78:1444-1445); 메탈로티오닌 유전자로부터의 프로모터 및 조절 시퀀스들(프린스터(Prinster)와 그의 동료들의 1982, Nature 296:39-42); 및 베타-락타마아제 프로모터(beta-lactamase promoter) 등과 같은 원핵 프로모터들(빌라-카마로프(Villa-Kamaroff)와 그의 동료들의 1978, Proc. Natl. Acad. Sci. U.S.A. 75:3727-3731); 또는 tac 프로모터(드보어와 그의 동료들의 1983, Proc. Natl. Acad. Sci. U.S.A. 80:21-25)들이 포함되나, 이들에 제한되는 것은 아니다. 조직 특이성을 나타내고 그리고 이식유전자를 갖는 동물들에서 활용되는 하기의 동물 전사 제어 영역들이 또한 흥미의 대상들이다: 췌장의 선방세포들 내에서 활성인 엘라스타아제 I 유전자(스위프트(Swift)와 그의 동료들의 1984, Cell 38:639-646; 오르니츠(Ornitz)와 그의 동료들의 1986, Cold Spring Harbor Symp. Quant. Biol. 50:399-409; 맥도널드의 1987, Hepatology 7:425-515); 췌장 베타세포들 내에서 활성인 인슐린 유전자 제어 영역(하나한(Hanahan)의 1985, Nature 315:115-122); 임파구들 내에서 활성인 면역글로블린 유전자 제어 영역(그로스체들(Grosschedl)과 그의 동료들의 1984, Cell 38:647-658; 아담스와 그의 동료들의 1985, Nature 318:533-538; 알렉산더와 그의 동료들의 1987, Mol. Cell. Biol. 7:1436-1444); 고환, 유방, 림포이드 및 비만세포들 내에서 활성인 생쥐 유선종양 바이러스 제어 영역(레더(Leder)와 그의 동료들의 1986, Cell 45:485-495); 간 내에서 활성인 알부민 유전자 제어 영역(핀커트(Pinkert)와 그의 동료들의 1987, Genes and Devel. 1:268-276); 간 내에서 활성인 알파-페토-단백질 유전자 제어 영역(크룸라우프(Krumlauf)와 그의 동료들의 1985, Mol. Cell Biol. 5:1639-1648; 햄머와 그의 동료들의 1987, Science 253:53-58); 간 내에서 활성인 알파 1-안티트립신 유전자 제어 영역(켈시(Kelsey)와 그의 동료들의 1987, Genes and Devel. 1:161-171); 골수 세포들 내에서 활성인 베타-글로빈 유전자 제어 영역(모그램(Mogram)과 그의 동료들의 1985, Nature 311:338-340; 콜리아스와 그의 동료들의 1986, Cell 46:89-94); 뇌내의 희돌기교세포들 내에서 활성인 수초기본단백질 유전자 제어 영역(리드헤드(Readhead)와 그의 동료들의 1987, Cell 48:703-712); 골격근 내에서 활성인 미오신 경쇄-2 유전자 제어 영역(사니(Sani)의 1985, Nature 314:283-286); 및 시상하부 내에서 활성인 성선자극 방출 호르몬 유전자 제어 영역(메이슨과 그의 동료들의 1986, Science 234:1372-1378).
인핸서 시퀀스가 상기 벡터 내로 삽입되어 보다 높은 진핵세포들에 의하여 인간 c-fms 항원 결합 단백질을 포함하는 경쇄 또는 중쇄를 인코딩하는 DNA의 전사를 증가시킬 수 있다. 인핸서들은 상기 프로모터에 작용하여 전사를 증가시키는, 길이에 있어서 대략 10 내지 300bp인 DNA의 시스-작용 요소(cis-acting elements)들이다. 인핸서들은 상대적으로 배향 및 위치 독립적이며, 상기 전사단위에 대해 5' 및 3' 둘 다의 위치들에서 발견된다. 포유동물 유전자들로부터 획득가능한 여러 인핸서 시퀀스들이 공지되어 있다(예를 들면, 글로빈, 엘라스타아제, 알부민, 알파-페토-단백질 및 인슐린). 그러나, 전형적으로, 바이러스로부터의 인핸서가 사용된다. 당해 기술분야에서 공지된 상기 SV40 인핸서, 상기 거대세포바이러스 초기 프로모터 인핸서, 상기 폴리오마 인핸서 및 아데노바이러스 인핸서들은 진핵 프로모터들의 활성화를 위한 예시적인 인핸싱 요소들이다. 비록 인핸서가 벡터 내에서 코딩 시퀀스에 대하여 5' 또는 3' 둘 중의 어느 하나에 위치될 수 있기는 하나, 전형적으로는 상기 프로모터로부터 5' 사이트에 위치된다. 적절한 자연의 또는 이종기원의 신호 시퀀스를 인코딩하는 시퀀스(리더 시퀀스 또는 신호 펩티드)는 발현 벡터 내로 내포되어 상기 항체의 세포외 분비를 촉진할 수 있다. 신호 펩티드 또는 리더의 선택은 상기 항체가 생산되어야 할 숙주세포들의 형태에 의존적이며, 이종기원의 신호 시퀀스는 상기 자연의 신호 시퀀스를 대체할 수 있다. 포유동물 세포들 내에서 기능적인 신호 펩티드들의 예들에는 다음의 것들이 포함된다: 미합중국 특허 제4,965,195호에 기술된 인터류킨-7(IL-7)에 대한 신호 시퀀스; 코스만(Cosman)과 그의 동료들의 1984, Nature 312:768에 기술된 인터류킨-2 수용체에 대한 신호 시퀀스; 유럽특허 제0367 566호에 기술된 인터류킨-4 수용체 신호 펩티드; 미합중국 특허 제4,968,607호에 기술된 I형 인터류킨-1 수용체 신호 펩티드; 유럽특허 제0 460 846호에 기술된 II형 인터류킨-1 수용체 신호 펩티드.
제공되는 상기 발현 벡터들은 통상적으로 획득가능한 벡터로의 개시 벡터로부터 구축될 수 있다. 이러한 벡터들은 원하는 측면서열의 전부를 포함하거나 또는 포함하지 않을 수 있다. 본 명세서에서 기술되는 상기 측면서열들의 하나 또는 그 이상이 상기 벡터 내에 이미 존재하지 않는 경우, 이들은 개별적으로 수득되고 그리고 상기 벡터 내로 결착될 수 있다. 상기 측면시퀀스들 각각을 얻기 위한 방법들은 당해 기술분야에서 숙련된 자들에게는 잘 알려져 있다.
상기 벡터가 구축되고 그리고 경쇄, 중쇄, 또는 c-fms 항원 결합 시퀀스를 포함하는 경쇄와 중쇄를 인코딩하는 핵산분자가 상기 벡터의 적절한 사이트 내로 삽입된 후, 완전한 벡터가 증폭 및/또는 폴리펩티드 발현을 위한 적절한 숙주세포 내로 삽입될 수 있다. 항원-결합 단백질을 위한 발현 벡터의 선택된 숙주세포 내로의 형질전환은 형질변환, 감염, 인산칼슘 공-침전, 전기천공, 미세주사, 리포펙션(lipofection), DEAE-덱스트란 매개 형질변환 또는 다른 공지의 기술들을 포함하여 공지의 방법들에 의해 수행될 수 있다. 선택되는 방법은 부분적으로는 사용되어야 할 숙주세포의 형태에 기능적일 것이다. 이들 방법들 및 다른 적절한 방법들이 숙련된 기술자들에게 잘 알려져 있으며, 예를 들면, 샘브룩과 그의 동료들의 위의 2001 문헌에 규정되어 있다.
적절한 조건들 하에서 배양되는 경우, 숙주세포는 대체로 상기 배지로부터 수집될 수 있거나(상기 숙주세포가 이를 상기 배지 내로 분비하는 경우) 또는 이를 생산하는 상기 숙주세포로부터 직접적으로 수집될 수 있는(분비되지 않는 경우) 항원 결합 단백질을 합성한다. 적절한 숙주세포의 선택은 원하는 발현 수준들, 활성에 대하여 바람직하거나 또는 필요한 펩티드 변형들(글리코실화 또는 인산화 등과 같은) 및 생물학적으로 활성인 분자 내로의 중첩의 용이화 등과 같은 다양한 인자들에 의존적일 수 있다.
발현을 위한 숙주들로서 획득가능한 포유동물 세포주들은 당해 기술분야에서 잘 알려져 있으며, 차이니스 햄스터 난소(CHO) 세포, 헬라세포, 베이비햄스터 신장(BHK) 세포, 원숭이 신장 세포(COS), 인간 원발성 간암 세포(예를 들면, Hep G2) 및 다수의 다른 세포주들을 포함하나 이들에 제한되지 않는 균주기탁 및 배포협회(ATCC ; American Type Culture Collection)로부터 획득가능한 불멸화된 세포주들이 포함되나, 이들에 제한되는 것은 아니다. 특정의 구체예들에 있어서, 세포주들은 어느 세포주들이 높은 발현수준들을 가지고 그리고 c-fms 결합 특성들을 갖는 항원 결합 단백질들을 구성적으로 생산하는 지를 결정하는 것을 통하여 선택될 수 있다. 다른 구체예에 있어서, 그 자체의 항체를 만들지는 않으나 이종기원의 항체를 만들고 그리고 분비하는 능력을 가진 상기 B세포 계통으로부터의 세포주가 선택될 수 있다.
진단 및
치료적인
목적들을 위한 인간 C-
fms
항원 결합 단백질들의 용도
항원 결합 단백질들은 생물학적 샘플들 내의 c-fms를 검출하고 그리고 c-fms를 생산하는 세포들 또는 조직들을 동정하는 데 유용하다. 예를 들면, 상기 c-fms 항원 결합 단백질들은, 진단분석, 예를 들면, 조직 또는 세포 내에서 발현된 c-fms를 검출 및/또는 정량하기 위한 결합분석에서 사용될 수 있다. c-fms에 특이적으로 결합하는 항원 결합 단백질들은 또한 치료를 요하는 환자 내에서 c-fms에 연관된 질병들의 치료에 사용될 수 있다. 게다가, c-fms 항원 결합 단백질들은 c-fms가 그의 리간드 CSF-1과 함께 착체를 형성하는 것을 저해하고 그에 의하여 세포 또는 조직 내에서의 c-fms의 생물학적 활성을 조정하는 데 사용될 수 있다. 조정될 수 있는 활성들의 예들에는 c-fms의 자가인산화의 저해, 단핵구 주화성의 감소, 단핵구 이동의 저해, 종양 또는 감염된 조직 내에서의 종양-연관 마크로파지의 축적의 저해 및/또는 신생혈관형성의 저해들이 포함되나, 이들에 제한되는 것은 아니다. 따라서 c-fms에 결합하는 항원 결합 단백질들은 다른 결합 화합물들과의 상호작용을 조정하거나 및/또는 차단할 수 있으며, 마찬가지로 c-fms와 연관되는 질병들을 완화시키는 치료적인 용도를 가질 수 있다.
징후들
많은 종양 세포들은 CSF-1을 분비하고, 이는 차례로 동족수용체 c-fms를 통하여 단핵구/마크로파지 세포들을 유인하고, 그의 생존을 촉진하고 그리고 활성화한다. 인간 종양들에 있어서 CSF-1의 수준은 이들 종양들 내에 존재하는 종양 연관 마크로파지의 수와 정의 관계로 연관되는 것이 밝혀졌다(머도크(Murdoch)와 그의 동료들의 2004, Blood 104:2224-2234). 많은 연구들이 여러 형태들의 암을 앓는 환자들에 있어서 높은 TAM 수들이 감소된 환자의 생존과 관련되어 있음을 보여주고 있다. 최근의 연구들은 종양 세포들 내의 자가분비 루프(autocrine loop)의 존재를 나타내고 있다. 다른 연구들은 c-fms가 여러 염증성 질환들에서 일정한 역할을 하는 것을 나타내고 있다. 따라서, 본 명세서에서 제공되는 상기 인간 c-fms 항원-결합 단백질들에 의한 c-fms-CSF-1 시그널링의 조절이 c-fms에 연관되는 생물학적 반응들 중의 적어도 하나를 저해하거나, 간섭하거나 또는 조절할 수 있으며, 또한 마찬가지로 c-fms-연관 질병들 또는 상태들의 영향들을 완화하는 데 유용하다. 본 명세서에서 제공되는 C-fms 결합 단백질들은 또한 이러한 질병들 또는 상태들의 진단, 예방 또는 치료에 사용될 수 있다.
인간 c-fms와 연관되는 질병 또는 상태에는 환자에서 그의 개시가 적어도 부분적으로는 cfms의 CSF-1 리간드 및/또는 IL-34와의 상호작용에 의해 야기되는 질병 또는 상태가 포함된다. 상기 질병 또는 상태의 심각성은 또한 c-fms의 CSF-1 및/또는 IL-34와의 상호작용에 의해 증가되거나 또는 감소될 수 있다. 상기 항원 결합 단백질들로 치료될 수 있는 질병들 및 상태들의 예들에는 여러 암들, 염증성 질병들 및 골질환들이 포함된다. 상기 항원 결합 단백질들은 또한 암의 전이 및 암의 뼈로의 전이와 연관된 골용해를 치료하거나 또는 예방하는 데 사용될 수 있다. TAMs의 높은 수준은 유방암(추추이(Tsutsui)와 그의 동료들의 2005, Oncol. Rep. 14:425-431; 리크(Leek)와 그의 동료들의 1999, Br. J. Cancer 79:991-995; 리크와 해리스(Leek and Harris)의 2002, J. Mammary Gland Biol and Neoplasia 7:177- 189), 전립선암(리스브란트(Lissbrant)와 그의 동료들의 2000, Int. J. Oncol. 17:445-451), 자궁내막암(오노(Ohno)와 그의 동료들의 2004, Anticancer Res. 24:3335-3342), 방광암(하나다(Hanada)와 그의 동료들의 2000, Int. J. Urol 7:263-269), 신장암(하마다(Hamada)와 그의 동료들의 2002, Anticancer Res. 22:4281-4284), 식도암(루이스와 폴라르드(Lewis and Pollard)의 2006, Cancer Res. 66(2):606-612), 편평상피세포암(코이데(Koide)와 그의 동료들의 2004, Am. J. Gastroenterol. 99:1667-1674), 포도막흑색종(마키티에(Makitie)와 그의 동료들의 2001, Invest. Ophthalmol. Vis. Sci. 42:1414-1421), 여포성림프종(follicular lymphoma ; 파린하(Farinha)와 그의 동료들의 2005, Blood l06:2169-2174), 신장 및 경부암(키르마(Kirma)와 그의 동료들의 2007, Cancer Res 67:1918-1926)들을 포함하여 다양한 암들에서의 종양 성장과 연관된다. 유방암, 전립선암, 자궁내막암, 방광암, 신장암, 식도암, 편평상피세포암, 포도막흑색종, 여포성림프종 및 난소암들의 경우들에 있어서, TAMs의 높은 수준들은 또한 감소된 환자 생존율을 나타내고 있다. 따라서, 본 명세서에서 제공되는 상기 c-fms 항원 결합 단백질들은 종양 내에서의 상기 TAMs의 원기회복을 저해하고 그리고 생존과 기능을 감소시키는 데 사용될 수 있으며, 따라서 종양 성장에 부의 관계로 영향을 주며, 환자 생존율을 증가시킨다.
치료될 수 있는 다른 암들에는 고형암들 대체로 폐암(lung cancer), 난소암(ovarian cancer), 결장암(colorectal cancer), 뇌(brain), 췌장(pancreatic), 두부(head), 목(neck), 간(liver), 백혈병(leukemia), 림프종(lymphoma) 및 호지킨씨병(Hodgkin's disease), 다발성 골수종(multiple myeloma ; 파린하와 그의 동료들의 2005, Blood 106:2169-2174), 골수종(melanoma), 위암(gastric cancer), 성상세포암(astrocytic cancer), 위장 및 폐의 선암(stomach and pulmonary adenocarcinoma. 이시가미(Ishigami)와 그의 동료들의 2003, Anticancer Research 23:4079-4083; 카루소와 그의 동료들의 1999, Modern Pathology 12:386-390; 위트처(Witcher)와 그의 동료들의 2004, Research Support 104:3335-3342; 하란-게라(Haran-Ghera)와 그의 동료들의 1997, Blood 89:2537-2545; 후세인(Hussein)과 그의 동료들의 2006 International Journal of Experimental Pathology 87:163-76; 라우와 그의 동료들의 2006 British Journal of Cancer. 94:1496-1503, 레웅(Leung)과 그의 동료들의 1997, Acta Neuropathologica. 93:518-527, 기라우도(Giraudo)와 그의 동료들의 2004, Journal of Clinical Investigation 114:623-633; 키르마(Kirma)와 그의 동료들의 2007, Cancer Research 67:1918-26, 반 라벤스와이(van Ravenswaay)와 그의 동료들의 1992, Laboratory Investigation 67: 166-174들이 포함되나, 이들에 제한되는 것은 아니다.
상기 항원 결합 단백질들은 또한 종양 성장, 진행 및/또는 전이를 저해하는 데 사용될 수 있다. 이러한 저해는 여러 방법들을 통하여 모니터링될 수 있다. 예를 들면, 저해는 감소된 종양 크기의 결과를 가져오거나 및/또는 상기 종양 내에서의 대사 활성(metabolic activity)를 감소시킬 수 있다. 이들 인자들 둘 다는 예를 들면 MRI 또는 PET 주사(PET scans)들에 의해 측정될 수 있다. 저해는 또한 괴사(necrosis), 종양 세포 사멸의 수준 및 상기 종양 내에서의 맥관질(vascularity)의 수준을 규명하기 위한 생체검사(biopsy)에 의해 모니터링될 수 있다. 전이의 정도 및 전이와 연관되는 골용해는 공지의 방법들을 사용하여 모니터링될 수 있다.
자가분비 루프의 존재에 대한 증거는 c-fms 활성의 저해가 마크로파지 연관 종양에 대하여 영향을 줄 수 있으나 또한 종양 세포들에 대하여도 영향을 줄 수 있다는 것을 나타내고 있다. 따라서, 하나의 구체예에 있어서, 자가분비 루프를 갖는 종양들이 1차적인 목표로서 표적화된다. 다른 구체예들에 있어서, TAMs 및 상기 종양들 둘 다가 결합된 효과를 위하여 표적화된다. 또 다른 구체예들에 있어서, 인접 분비(paracrine) 또는 자가분비와 인접 분비를 사용하는 종양들이 표적화된다.
본 명세서에서 제공되는 상기 인간 c-fms 항원 결합 단백질들은 특정의 구체예들에 있어서 단독으로 투여될 수 있으나, 또한 예를 들면 화학요법(chemotherapy), 방사선요법(radiation therapy) 또는 외과적 수술(surgery) 등과 같은 하나 또는 그 이상의 다른 암치료 옵션(options)들과 조합으로 사용될 수 있다. 화학요법제들과 함께 투여되는 경우, 상기 항원 결합 단백질은 상기 화학요법제 이전 또는 이후에 또는 동시에(예를 들면, 동일한 조성물의 일부로서) 투여될 수 있다.
제공되는 상기 항원 결합 단백질들과 조합으로 사용될 수 있는 화학요법 치료제들에는 메클로레타민(mechlorethamine), 사이클로포스파미드(cyclophosphamide), 이포스파미드(ifosfamide), 멜파란(melphalan) 및 클로람부실(chlorambucil) 등과 같은 질소 머스터드(nitrogen mustards)를 포함하는 알킬화제(alkylating agents)를 포함하는 항-신생물 약제(anti-neoplastic agents); 카무스틴(carmustine ; BCNU), 로무스틴(lomustine ; CCNU) 및 세무스틴(semustine ; methyl-CCNU) 등과 같은 니트로소우레아(nitrosoureas); 테모달™(Temodal™ ; 테모졸아미드), 트리에틸렌멜라민(TEM), 트리에틸렌티오포스포르아미드(티오테파), 헥사메틸멜라민(HMM, 알트레타민) 등과 같은 에틸렌이민/메틸멜라민(ethylenimines/methylmelamine); 부설판(busulfan) 등과 같은 알킬설포네이트(alkyl sulfonates); 다카바진(dacarbazine ; DTIC) 등과 같은 트리아진(triazines); 메토트렉세이트 및 트리메트렉세이트 등과 같은 엽산 유사체들(folic acid analogs), 5-플루오로우라실(5FU), 플루오로데옥시우리딘, 겜시타빈, 시토신 아라비노시드(AraC, 시타라빈), 5-아자시티딘, 2,2'-디플루오로데옥시시티딘 등과 같은 피리미딘 유사체들, 6-머캅토퓨린, 6-티오구아닌, 아자티오프린, T-데옥시코포르마이신(펜토스타틴), 에리트로히드록시노닐아데닌(EHNA), 플루다라빈포스페이트 및 2-클로로데옥시아데노신(클라드리빈, 2-CdA) 등과 같은 퓨린 유사체들을 포함하는 대사길항제들(antimetabolites); 파클리탁셀 등과 같은 항유사분열약물(antimitotic drugs), 빈블라스틴(VLB), 빈크리스틴 및 비노렐빈, 탁소테레, 에스트라무스틴 및 에스트라무스틴포스페이트들을 포함하는 빈카알칼로이드들(vinca alkaloids)을 포함하는 자연의 생성물들; 에토포시드 및 테니포시드 등과 같은 피포도필로톡신(pipodophylotoxins); 악티모마이신 D, 다우노마이신(루비도마이신), 독소루비신, 미토크산트론, 이다루비신, 블레오마이신, 플리카마이신(미트라마이신), 미토마이신 C 및 악티노마이신 등과 같은 항생제들; 엘-아스파라기나아제 등과 같은 효소들; 알파-인터페론, IL-2, G-CSF 및 GM-CSF 등과 같은 생물학적 반응 변형제들(biological response modifiers); 시스플라틴 및 카보플라틴 등과 같은 백금 배위 착물들, 미토크산트론 등과 같은 안트라센디온, 히드록시우레아 등과 같은 치환된 요소, N-메틸히드라진(MIH) 및 프로카바진을 포함하는 메틸히드라진 유도체들, 미토탄(o,p-DDD) 및 아미노글루테티미드 등과 같은 부신피질 억제제들(adrenocortical suppressants)을 포함하는 기타 약품들; 프레드니손 및 등가물들, 덱사메타손 및 아미노글루테티미드 등과 같은 부신피질스테로이드 길항제들(adrenocorticosteroid antagonists)을 포함하는 호르몬 및 길항제들; 겜자르™(Gemzar™ ; 겜시타빈), 히드록시프로게스테론 카프로에이트, 메드록시프로게스테론아세테이트 및 메게스트롤아세테이트 등과 같은 프로게스틴(progestin); 디에틸스틸베스트롤 및 에티닐에스트라디올 등가물 등과 같은 에스트로겐; 타목시펜 등과 같은 항에스트로겐(antiestrogen); 테스토스테론프로피오네이트 및 플루옥시메스테론/등가물들을 포함하는 안드로겐들(androgens); 플루타미드, 고나도트로핀-방출 호르몬 유사체들 및 류프롤리드 등과 같은 항안드로겐들(antiandrogens); 및 플루타미드 등과 같은 비-스테로이드성 항안드로겐들이 포함되나, 이들에 제한되는 것은 아니다. 히스톤 디아세틸라아제 저해제들(histone deacetylase inhibitors), 디메틸화 약품들(demethylating agents ; 예를 들면, 비다자(Vidaza))들 및 전사 억제제의 방출(release of transcriptional repression ; ATRA) 요법들을 포함하는 후성학적 메카니즘(epigenetic mechanism)을 목표하는 요법들이 또한 상기 항원 결합 단백질들과 조합될 수 있다.
항원 결합 단백질로 관리될 수 있는 암요법들에는 또한 목표화된 요법(targeted therapies)들이 포함되나, 이들에 제한되는 것은 아니다. 목표화된 요법들의 예들에는 치료적인 항체들의 사용이 포함되나, 이들에 제한되는 것은 아니다. 예시적인 치료적인 항체들에는 생쥐 항체들, 생쥐-인간 키메라 항체들, CDR-분지화된 항체들, 인간화된 항체들 및 완전한 인간 항체들 그리고 항체 라이브러리들을 스크리닝하는 것에 의하여 선택되는 합성 항체들이 포함되나, 이들에 제한되는 것은 아니다. 예시적인 항체들에는 세포 표면 단백질들 Her2, CDC20, CDC33, 뮤신-형 당단백질 및 종양 세포들 상에 존재하는 표피성장인자 수용체(epidermal growth factor receptor ; EGFR)에 결합하고 그리고 선택적으로 이들 단백질들을 디스플레이하는 종양 세포들 상에 세포증식억제 및/또는 세포독성 효과를 유도하는 것들이 포함되나, 이들에 제한되는 것은 아니다. 예시적인 항체들은 또한 유방암 및 암의 다른 형태들을 치료하는 데 사용될 수 있는 허셉틴™(HERCEPTIN™ ; 트라스트주마브(trastuzumab)) 및 비-호지킨스 림프종과 암의 다른 형태들을 치료하는 데 사용될 수 있는 리툭산™(RITUXAN™ ; 리툭시마브(rituximab)), 제발린™(ZEVAL1N™ ; 이브리투모마브 티욱세탄(ibritumomab tiuxetan)), 글리벡™(GLEEVEC™) 및 림포사이드™(LYMPHOCIDE™ ; 에프라투주마브(epratuzumab))들이 포함된다. 특정의 예시적인 항체들에는 또한 에르비툭스™(ERBITUX™ ; EMC-C225); 에르티놀리브(Iressa); 벡사르™(BEXXAR™ ; iodine 131 tositumomab); KDR(키나아제 도메인 수용체) 저해제들; 항VEGF 항체들 및 길항제들(예를 들면, 아바스틴™(Avastin™) 및 VEGAF-TRAP); 항VEGF 수용체 항체들 및 항원 결합 영역들; 항-Ang-1 및 Ang-2 항체들 및 항원 결합 영역들; Tie-2 및 다른 Ang-1 및 Ang-2 수용체들에 대한 항체들; Tie-2 리간드들; Tie-2 키나아제 억제제들에 대한 항체들; inhibitors of Hif-la의 저해제들 및 캄파쓰™(Campath™ ; 알렘투주마브)들이 포함된다. 특정의 구체예들에 있어서, 암요법제들은 TNF-연관 폴리펩티드 TRAIL을 포함하나, 이들에 제한되지 않는, 종양 세포들 내에서 세포사멸(apoptosis)를 선택적으로 유도하는 폴리펩티드들이다.
화학요법제들의 부가의 특정의 예들에는 탁솔, 탁센(예를 들면, 도세탁셀 및 탁소테레), 변형된 파클리탁셀(예를 들면, 아브락센 및 오팍시오) 독소루비신, 아바스틴®(Avastin®), 수텐트(Sutent), 넥사바르(Nexavar) 및 다른 다중표적항암제(multikinase inhibitors), 시스플라틴과 카보플라틴, 에토포시드, 겜시타빈 및 빈블라스틴들이 포함된다. MAPK 경로 저해제들(예를 들면, ERK, JNK 및 p38의 저해제들), PB키나아제/AKT 저해제들 및 Pim 저해제들들을 포함하나, 이들에 제한되지 않는 다른 키나아제들의 저해제들이 또한 상기 항원 결합 단백질들과 조합으로 사용될 수 있다. 다른 저해제들에는 Hsp90 저해제들, 프로테아좀 저해제들(proteasome inhibitors ; 예를 들면, 벨케이드(Velcade)) 및 트리세녹스(Trisenox) 등과 같은 작용 저해제(action inhibitors)들의 다중 메카니즘(multiple mechanism) 들이 포함된다.
특정의 구체예에 있어서, 본 명세서에서 제공되는 바와 같은 항원 결합 단백질은 신생혈관형성을 감소시키는 하나 또는 그 이상의 항-신생혈관형성약품(anti-angiogenic agents)들과 조합으로 사용된다. 이러한 약품들에는 IL-8 길항제들; 캄파쓰, B-FGF; FGF 길항제들; Tek 길항제들(세레티와 그의 동료들의 미합중국 특허공개공보 제2003/0162712호; 세레티와 그의 동료들의 미합중국 특허 제6,413,932호 및 동 제6,521,424호, 이들 각각은 본 명세서에서 모든 목적들에 대하여 참조로 인용함); 항-TWEAK 약품들(여기에는 항체들 및 항원 결합 단백질들이 포함되나, 이들에 제한되는 것은 아님); 가용성 TWEAK 수용체 길항제들(윌리의 미합중국 특허 제6,727,225호); 그의 리간드들에의 인테그린의 결합을 길항하는 ADAM 디스인테그린 도메인(ADAM distintegrin domain)(판슬로우(Fanslow)와 그의 동료들의 미합중국 특허공개공보 제2002/0042368호); 항-eph 수용체 및 항-에프린 항체들; 항원 결합 영역들, 또는 길항제들(미합중국 특허 제5,981,245호; 동 제5,728,813호; 동 제5,969,110호; 동 제6,596,852호; 동 제6,232,447호; 동 제6,057,124호 및 이들의 패밀리특허들); 아바스틴™(Avastin™) 또는 VEGF-TRAP™ 등과 같은 항-VEGF 약품들(예를 들면, VEGF 또는 가용성 VEFG 수용체들 또는 이들의 리간드 결합 영역들을 특이적으로 결합하는 항체들 또는 항원 결합 영역들)과 항-VEGF 수용체 약품들(예를 들면, 이들에 특이적으로 결합하는 항체들 또는 항원 결합 단백질들), 파니투무마브(panitumumab), 이레싸™(IRESSA™ ; gefitinib), 타르세프 에이™(TARCEV A™ ; erlotinib) 등과 같은 EGFR 저해 약품들(예를 들면, 이들에 특이적으로 결합하는 항체들 또는 항원 결합 영역들), 항-Ang-1 및 항-Ang-2 약품들(예를 들면, 이들 또는 이들의 수용체들, 예를 들면, Tie-2/TEK에 특이적으로 결합하는 항체들 또는 항원 결합 영역들) 및 항-Tie-2 키나아제 저해약품들(예를 들면, 특이적으로 결합하고 그리고 간세포 성장 인자(hepatocyte growth factor ; HGF, 또한 스캐터 인자(Scatter Factor)로도 알려진)의 길항제들 등과 같은 성장 인자들의 활성을 저해하는 항체들 또는 항원 결합 영역들) 및 그의 수용체 "c-met"에 특이적으로 결합하는 항체들 또는 항원 결합 영역들; 항-PDGF-BB 길항제들; PDGF-BB 리간드들에 대한 항체들 및 항원 결합 영역들; 및 PDGFR 키나아제 저해제들이 포함되나, 이들에 제한되는 것은 아니다.
항원 결합 단백질과 조합하여 사용될 수 있는 다른 항-길항제들에는 MMP-2(매트릭스-메탈로프로테이나아제 2 ; matrix- metalloproteinase 2) 저해제들, MMP-9(매트릭스-메탈로프로테이나아제 9) 및 COX-II(사이클로옥시게나아제 II) 저해제들이 포함된다. 유용한 COX-II 저해제들의 예들에는 세레브렉스™(CELEBREX™ ; 셀레콕시브), 발데콕시브 및 로페콕시브들이 포함된다.
특정의 구체예들에 있어서, 암요법제들은 신생혈관형성 저해제들이다. 이러한 저해제들에는 SD-7784(미합중국, 화이자(Pfizer)); 실렝기타이드(독일, 머크(Merck KGaA), 유럽특허 제770622호); 페갑타니브옥타소듐(미합중국, 길리드 사이언시즈(Gilead Sciences)); 알파스타틴(영국, 바이오악타(BioActa)); M-PGA(미합중국, 셀젠(Celgene), 미합중국 등록특허 제5,712,291호); 일로마스타트(미합중국, 아리바(Arriva), 미합중국 등록특허 제5,892,112호); 세막사니브(semaxanib), (미합중국, 화이자, 미합중국 등록특허 제5,792,783호); 바탈라니브(vatalanib), (스위스, 노바티스(Novartis)); 2-메톡시에스트라디올(2-methoxyestradiol), (미합중국, 엔터메드(EntreMed)); 티엘씨 이엘엘-12(TLC ELL-12), (아일랜드, 엘란(Elan)); 아네코르타브(anecortave acetate), (미합중국, 알콘(Alcon)); 알파-디148 마브(alpha-D148 Mab), (미합중국, 암겐(Amgen)); 세프-7055(CEP-7055), (미합중국, 세팔론(Cephalon)); 항-Vn 마브(anti-Vn Mab), (네덜란드, 크루셀(Crucell)) 디에이씨:항신생혈관형성(DAC:antiangiogenic), (캐나다, 콘쥬켐(ConjuChem)); 안지오시딘(Angiocidin), (미합중국, 인카인 파마슈티칼(InKine Pharmaceutical)); 케이엠-2550(KM-2550), (일본, 교와학코(Kyowa Hakko)); 에스유-0879(SU-0879), (미합중국, 화이자); 씨지피-79787(CGP-79787), (스위스, 노바티스, 유럽특허 제970070호); 아르젠트 테크놀로지(ARGENT technology), (미합중국, 아리아드(Ariad)); 와이아이지에스알-스틸스(YIGSR-Stealth), (미합중국, 존슨 앤 존슨(Johnson & Johnson)); 피브리노겐-이 단편(fibrinogen-E fragment), (영국, 바이오악타(BioActa)); 신생혈관형성 저해제(angiogenesis inhibitor), (영국, 트리젠(Trigen)); 티비씨-1635(TBC-1635), (미합중국, 엔사이시브 파마슈티컬스(Encysive Pharmaceuticals)); 에스씨-236(SC-236), (미합중국, 화이자); 에이비티-567(ABT-567), (미합중국, 애보트(Abbott)); 메타스타틴(Metastatin), (미합중국, 엔터메드); 신생혈관형성 저해제, (스웨덴, 트리펩(Tripep)); 마스핀(maspin), (일본, 소세이(Sosei)); 2-메톡시에스트라디올(2-methoxyestradiol), (미합중국, 온콜로지 사이언시즈 코포레이션(Oncology Sciences Corporation)); 이알-68203-00(ER-68203-00), (미합중국, 이박스(IVAX)); 베네핀(Benefin), (미합중국, 레인 랩스(Lane Labs)); 티지-93(Tz-93), (일본, 츠무라(Tsumura)); 티에이엔-1120(TAN-1120), (일본, 다케다(Takeda)); 에프알-111142(FR-111142), (일본, 후지사와(Fujisawa), 일본특허 제02233610호); 플레이트렛 팩터 4(platelet factor 4), (미합중국, 레플리젠(RepliGen), 유럽특허 제407122호); 혈관내피세포 성장 인자 길항제(vascular endothelial growth factor antagonist), (덴마크, 보레안(Borean)); 암요법(cancer therapy), (미합중국, 유니버시티 오브 사우스 캐롤라이나(University of South Carolina)); 베바시주마브(피아이엔엔)((bevacizumab (pINN)), (미합중국, 제넨테크(Genentech)); 신생혈관형성 저해제들(미합중국, 수젠(SUGEN)); 엑스엘 784(XL 784), (미합중국, 엑셀릭시스(Exelixis)); 엑스엘 647(미합중국, 엑셀릭시스); 마브, 알파5베타3 인테그린, 2세대(MAb, alpha5beta3 integrin, second generation), (미합중국, 어플라이드 몰레큘러 에볼루션, 유에스에이 앤드 메드임뮨(Applied Molecular Evolution, USA and MedImmune)); 진테라피, 레티노파씨(gene therapy, retinopathy), (영국, 옥스포드 바이오메디카(Oxford BioMedica)); 엔자스타우린하이드로클로라이드 (유에스에이엔)(enzastaurin hydrochloride (USAN)), (미합중국, 릴리(Lilly)); 세프 7055(CEP 7055), (프랑스, 세팔론, 유에스에이 앤드 사노피-신디라보(Cephalon, USA and Sanofi-Synthelabo)); 비씨 1(BC 1), (이태리, 제노아 인스티튜트 오브 캔서 리서치(Genoa Institute of Cancer Research)); 신생혈관형성 저해제(오스트레일리아, 알케미아(Alchemia)); VEGF 길항제(VEGF anagonist), (미합중국, 레제네론(Regeneron)); 알비피아이 21 및 비피아이-유도 길항제(rBPI 21 and BPI-derived antiangiogenic), (미합중국, 소마(XOMA)); 피아이 88(PI 88), (오스트레일리아, 프로젠(Progen)); 실렌지타이드 (피아이엔엔)((cilengitide (pINN)), (독일, 머크(Merck KGaA); 독일, 뮈니크 테크니컬 유니버시티(Munich Technical University), 미합중국, 스크립스 클리닉 앤드 리서치 파운데이션(Scripps Clinic and Research Foundation)); 세툭시마브 (아이엔엔)(cetuximab (INN)), (프랑스, 아벤티스(Aventis)); 에이브이이 8062(AVE 8062), (일본, 아지노모토(Ajinomoto)); 에이에스 1404, (뉴질랜드, 캔서 리서치 라보레토리(Cancer Research Laboratory)); 에스지 292, (미합중국, 텔리오스(Telios)); 엔도스타틴(Endostatin), (미합중국, 보스톤 아동병원(Boston Childrens Hospital)); 에이티엔 161, (미합중국, 아테뉴온(Attenuon)); 안지오스타틴(ANGIOSTATIN), (미합중국, 보스톤 아동병원); 2-메톡시에스트라디올, (미합중국, 보스톤 아동병원); 지디 6474, (영국, 아스트라제네카(AstraZeneca)); 지디 6126, (영국, 안지오젠 파마슈티칼스(Angiogene Pharmaceuticals)); 피피아이 2458, (미합중국, 프래시스(Praecis)); 에이지디 9935, (영국, 아스트라제네카); 에이지디 2171, (영국, 아스트라제네카); 바탈라니브 (페아이엔엔), (독일, 노바티스, 스위처랜드 앤드 쉐링 아게(Switzerland and Schering AG)); 조직 인자 경로 저해제들, (미합중국, 엔터메드); 페갑타니브 (피아이엔엔), (미합중국, 길리드 사이언시즈); 크산토르라이졸(xanthorrhizol), (대한민국, 연세대학교); 유전자-기반, VEGF-2 백신(vaccine, gene-based, VEGF-2), (미합중국, 스크립스 클리닉 앤드 리서치 파운데이션); 에스피브이5.2, (캐나다, 수프라텍(Supratek)); 에스디엑스 103, (미합중국, 캘리포니아 대학교 샌디에고(University of California at San Diego)); 피엑스 478, (미합중국, 프로익스(ProIX)); 메타스타틴(METASTATIN), (미합중국, 엔터메드); 트로포닌 1(troponin 1), (미합중국 하버드 대학교); 에스유 6668, (미합중국, 수젠); 오엑스아이 4503, (미합중국 옥시젠(OXiGENE)); o-구아니딘(o-guanidines), (미합중국, 디멘셔널 파마슈티칼스(Dimensional Pharmaceuticals)); 모투포르아민 씨(motuporamine C), (캐나다, 브리티시 컬럼비아 유니버시티(British Columbia University)); 씨디피 791, (영국, 셀텍 그룹(Celltech Group)); 아티프리모드 (피아이엔엔)((atiprimod (PINN)), (영국, 글락소스미스클라인(GlaxoSmithKline)); 이 7820(E 7820), (일본, 아이사이(Eisai)); 씨와이씨 381, (미합중국, 하버드 대학교); 에이이 941, (캐나다, 애테르나(Aeterna)); 신생혈관형성 백신(vaccine, angiogenesis), (미합중국, 엔터메드); 유로키나아제 플라스미노겐 활성자 저해제(urokinase plasminogen activator inhibitor), (미합중국, 당드레온(Dendreon)); 오글루파니드 (피아이엔엔)(oglufanide (pINN)), (미합중국, 멜모트(Melmotte)); 에이취아이에프-1 알파 저해제(HIF-1 alfa inhibitors), (영국, 제노바(Xenova)); 씨이피 5214, (미합중국, 세팔론(Cephalon)); 베이 레스 2622(BAY RES 2622), (독일, 바이엘(Bayer)); 안지오시딘(Angiocidin), (미합중국, 인키네(InKine)); 에이6(A6), (미합중국, 앙스트롬(Angstrom)); 케이알 31372, (대한민국, 한국화학연구원); 지더블류 2286, (영국, 글락소스미스클라인); 이에이취티 0101, (프랑스, 엑손히트(ExonHit)); 씨피 868596, (미합중국, 화이자); 씨피 564959, (미합중국, 오에스아이(OSI)); 씨피 547632, (미합중국, 화이자); 786034, (영국, 글락소스미스클라인); 케이알엔 633, (일본, 기린맥주(Kirin Brewery)); 2-메톡시에스트라디올, 안구 내 약물전달 시스템(drug delivery system, intraocular, 2- methoxyestradiol), (미합중국, 엔터메드); 안지넥스(anginex), (네덜란드, 마스트릭트 유니버시티(Maastricht University) 및 미합중국, 미네소타 유니버시티); 에이비티 510, (미합중국, 애보트(Abbott)); 엠엘 993, (스위스, 노바티스); 브이이지아이, (미합중국, 프로테옴 테크(Proteom Tech)); 종양괴사인자-알파 저해제(tumor necrosis factor-alpha inhibitors), (미합중국, 국립노화연구소(National Institute on Aging)); 에스유 11248, (미합중국, 화이자 및 미합중국, 수젠); 에이비티 518, (미합중국, 애보트); 와이에이취아이16, (중국, 얀타이 롱창(Yantai Rongchang)); 에스-3에이피지, (미합중국, 보스톤 아동병원 및 미합중국, 엔터메드); 마브, 케이디알, (미합중국, 임클론 시스템즈(ImClone Systems)); 마브, 알파5 베타1(MAb, alpha5 betal), (미합중국, 프로테인 디자인(Protein Design)); 케이디알 키나아제 저해제(KDR kinase inhibitor), (영국, 셀텍 그룹 및 미합중국, 존슨 앤 존슨); 지에프비 116, (미합중국, 사우스 플로리다 유니버시티(South Florida University) 및 미합중국, 예일대학교); 씨에스 706, (일본, 산쿄(Sankyo)); 컴브레타스타틴 에이4 프로드러그(combretastatin A4 prodrug), (미합중국, 아리조나주립대학교); 콘드로이티나아제 에이씨(chondroitinase AC), (캐나다, 이벡스(IBEX)); 베이 레스 2690, (독일, 바이엘); 에이지엠 1470, (미합중국, 하버드 대학교, 일본, 다케다, 미합중국, 티에이피(TAP)); 에이지 13925, (미합중국, 아고우론(Agouron)); 테트라티오몰리브데이트(Tetrathiomolybdate), (미합중국, 유니버시티 오브 미시간)); 지씨에스 100, (미합중국, 웨인주립대학교) 씨브이 247, (영국, 아이비 메디칼(Ivy Medical)); 씨케이디 732, (대한민국, 종근당); 마브, 혈관내피세포 성장 인자, (영국, 제노바); 이르소글라딘 (아이엔엔)(irsogladine (INN)), (일본, 일본신약(Nippon Shinyaku)); 알지 13577, (프랑스, 아벤티스); 더블류엑스 360, (독일, 윌렉스(Wilex)); 스쿠알라민 (피아이엔엔)(squalamine (pINN)), (미합중국, 젠아에라(Genaera)); 알피아이 4610, (미합중국, 시그마(Sima)); 암요법, (오스트레일리아, 마리노바(Marinova)); 헤파리나아제 저해제(heparanase inhibitors), (이스라엘, 인사이트(InSight)); 케이엘 3106, (대한민국, 코오롱); 호노키올(Honokiol), (미합중국, 에모리 유니버시티(Emory University)); 지케이 씨디케이(ZK CDK), (독일, 쉐링 아게(Schering AG)); 지케이 안지오(ZK Angio), (독일, 쉐링 아게); 지케이 229561, (스위스, 노바티스 및 독일, 쉐링 아게); 엑스엠피 300, (미합중국, 소마); 브이지에이 1102, (일본, 타이쇼(Taisho)); 브이이지에프 수용체 조절자(VEGF receptor modulators), (미합중국, 파마코피아(Pharmacopeia)); 브이이-카드헤린-2 길항제(VE-cadherin-2 antagonists), (미합중국, 임클론 시스템즈); 바소스타틴(Vasostatin), (미합중국, 국립건강연구소(National Institutes of Health)); Flk-1 백신(vaccine, Flk-1), (미합중국, 임클론 시스템즈); 티지 93, (일본, 츠무라(Tsumura)); 툼스타틴(TumStatin), (미합중국, 베스 이스라엘 호스피탈(Beth Israel Hospital)); 끝이 절단된 가용성 에프엘티 1(혈관내피세포 성장 인자 수용체 1)(truncated soluble FLT 1 (vascular endothelial growth factor receptor 1)), (미합중국, 머크 앤 컴퍼니(Merck & Co)); 티에-2 리간드(Tie-2 ligands), (미합중국, 리제네론(Regeneron)); 트롬보스폰딘 1 저해제(thrombospondin 1 inhibitor), (미합중국, 알레게니 헬스, 에듀케이션 앤드 리서치 파운데이션(Allegheny Health, Education and Research Foundation)); 2-벤젠설폰아미드(2-Benzenesulfonamide), 4-(5-(4-클로로페닐)-3-(트리플루오로메틸)-lH-피라졸-l-일)(4-(5-(4-chlorophenyl)-3-(trifluoromethyl)-lH-pyrazol-l-yl)-); 아리바(Arriva); 및 C-MeL AVE 8062
(2S)-2-아미노-3-히드록시-N-[2-메톡시-5-[(1Z)-2-(3,4,5-트리-메톡시페닐)에테닐]페닐]프로판아미드 모노하이드로클로라이드); 메텔리무마브 (피아이엔엔)(metelimumab (pINN))(면역글로블린 지4, 항-(인간 형질전환 성장인자.베타1.(인간 모노클로날 씨에이티 192.감마.4-쇄))(metelimumab (pINN)(immunoglobulin G4, anti-(human transforming growth factor.beta.l (human monoclonal CAT 192.gamma.4-chain)), 인간 모노클로날 씨에이티 192.카파.-쇄 이량체를 갖는 이황화물(disulfide with human monoclonal CAT 192.kappa.-chain dimer)); Flt3 리간드; CD40 리간드; 인터루킨-2; 인터루킨-12; 4-1BB 리간드; 항-4-IBB 항체들; TNFR/Fc를 포함하는 TNF 길항제들 및 TNF 수용체 길항제들, TWEAK-R/Fc를 포함하는 TWEAK 길항제들 및 TWEAK-R 길항제들; 트레일(TRAIL); 항-VEGF 항체들을 포함하는 VEGF 길항제들; VEGF 수용체(VEGF-Rl 및 VEGF-R2 포함, Fltl 및 Flkl 또는 KDR로도 알려짐) 길항제들; CDl48(DEP-1, ECRTP, 및 PTPRJ들로도 알려짐, 여기에 임의의 목적에 대하여 참조로 인용하는 다카하시(Takahashi)와 그의 동료들의 J. Am. Soc. Nephrol. 10:213545 (1999)를 참조하시오) 항진제(agonists); 트롬보스폰딘 1 저해제(thrombospondin 1 inhibitor) 및 Tie-2 또는 Tie-2 리간드(Ang-2 등과 같은)들의 어느 하나 또는 둘 다의 저해제들이 포함되나, 이들에 제한되는 것은 아니다. 미합중국 특허출원 제20030124129호(국제특허출원 제WO03/030833호에 대응) 및 미합중국 특허 제6,166,185호들에 기술된 항-Ang-2 항체들을 포함하여 Ang-2의 다수의 저해제들이 당해 기술분야에서 공지되어 있으며, 상기 문헌들의 내용들을 그들 전체로 본 명세서에 참조로 인용한다. 게다가, Ang-2 펩티바디(Ang-2 peptibodies)들이 당해 기술분야에서 공지되어 있으며, 예를 들면, 미합중국 특허공개공보 제20030229023호(국제특허출원 제WO03/057134호에 대응) 및 동 제20030236193호에서 발견될 수 있으며, 상기 문헌들의 내용들을 그들 전체로 본 명세서에서 참조로 인용한다.
특정의 암치료제(cancer therapy agents)들에는 탈리도미드 및 탈리도미드 유사체들 (N-(2,6-디옥소-3-피페리딜)프탈이미드); 테코갈란소듐(tecogalan sodium)(황산염처리된 폴리사카라이드 펩티도글리칸); 탄 1120(TAN 1120) (S-아세틸-7,8,9,10-테트라하이드로-6,8,11-트리하이드록시-l-메톡시-10-[[옥타하이드로-5-하이드록시-2-(2-하이드록시프로필)-4,10-디메틸피라노[3,4-d]-l,3,6-디옥사조신-8-일]옥시]-5,12-n나프타세네디온); 수라디스타(suradista) (7,7'-[카르보닐비스[이미노(1-메틸-1H-피롤-4,2-디일)카르보닐이미노(1-메틸-1H-피롤-4,2-디일)카르보닐이미노]]비스1,3-나프탈렌디설폰산 테트라소듐염); 에스유 302(SU 302); 에스유 1498 ((E)-2-시아노-3-[4-하이드록시-3,5-비스(1-메틸에틸)페닐]-N-(3-페닐프로필)-2-프로펜아미드); 에스유 1433 (4-(6,7-디메틸-2-퀴녹살리닐)-l-2-벤젠디올); 에스티 1514(ST 1514); 에스알 25989(SR 25989); 가용성 Tie-2; 세름 유도체들(SERM derivatives), 파모스(Pharmos); 세막사니브 (피아이엔엔)(semaxanib (pINN)) (3-[(3,5-디메틸-lH-피롤-2-일)메틸렌]-l,3-디하이드로-2H-인돌-2-온); 에스 836(S 836); 알지 8803(RG 8803); 레스틴(RESTIN); 알 440(R 440) (3-(l-메틸-lH-인돌-3-일)-4-(l-메틸-6-니트로-lH-인돌-3-일)-lH-피롤-2,5-디온); 알 123942 (l-[6-(l,2,4-티아디아졸-5-일)-3-피리다지닐]-N-[3-(트리플루오로메틸)페닐]-4-피페리딘아민); 프롤일 하이드록실라에제 저해제(prolyl hydroxylase inhibitor); 진행 강화 유전자들(progression elevated genes); 프리모마스태트 (아이엔엔)(prinomastat (INN)) ((S)-2,2-디메틸-4-[[p-(4-피리딜옥시)페닐]설포닐]-3-티오몰포린카르보하이드록사민산); 엔브이 1030; 엔엠 3 (8-하이드록시-6-메톡시-알파-메틸-l-옥소-lH-2-벤조피란-3-아세트산); 엔에프 681; 엔에프 050; 엠아이지; 메쓰 2(METH 2); 메쓰 1; 마나산틴 비(manassantin B) (알파-[l-[4-[5-[4-[2-(3,4-디메톡시페닐)-2-하이드록시-l-메틸에톡시]-3-m-에톡시페닐]테트라하이드로-3,4-디메틸-2-푸라닐]-2-메톡시페녹시]에틸]-l-3-벤조디옥솔-5-메탄올); KDR 모노클로날 항체; 알파5베타3 인테그린 모노클로날 항체; 엘와이 290293 (2-아미노-4-(3-피리디닐)-4H-나프토[l,2-b]-피란-3-카보니트릴); 케이피 0201448; 케이엠 2550; 인테그린-특이성 펩티드; 아이엔지엔 401; 지와이케이아이 66475; 지와이케이아이 66462; 그린스타틴 (101-354-플라스미노겐(인간)); 류마티스성관절염, 전립선암, 난소암, 신경아교종용 유전자치료법, 엔도스타틴, 결장암, ATF BTPI, 항신생혈관형성 유전자, 신생혈관형성 저해제 또는 신생혈관형성; 젤라틴가수분해효소 저해제, 에프알 111142 (4,5-디하이드록시-2-헥사논산 5-메톡시-4-[2-메틸-3-(3-메틸-2-부테닐)옥시라닐]-l-옥사피로[2.5]옥트-6-일에스터); 포르페니멕스 (피아이엔엔)(forfenimex (PINN)) (S)-알파-아미노-3-하이드록시-4-(하이드록시메틸)벤젠아세트산); 피브로넥틴 길항제 (1-아세틸-L-프롤릴-L-히스티딜-L-세릴-L-시스테이닐-L-아스파르타미드); 섬유아세포 성장 인자 수용체 저해제; 섬유아세포 성장 인자 길항제; 에프씨이 27164 (7,7'-[카르보닐비스[이미노(l-메틸-lH-피롤-4,2-디일)카르보닐이미노(l-메틸-lH-피롤-4,2-디일)카르보닐이미노]-]비스-l,3,5-나프탈렌트리설폰산 헥사소듐염); 에프씨이 26752 (8,8'-[카르보닐비스[이미노(l-메틸-lH-피롤-4,2-디일)카르보닐이미노(l-메틸-lH-피롤-4,2-디일)카르보닐이미노]]비스-l,3,6-나프탈렌트리설폰산); 내피세포의 단핵구 활성화 폴리펩티드 II(endothelial monocyte activating polypeptide II); VEGFR 항감작 올리고뉴클레오티드; 항-신생혈관생성 및 영양 인자들; ANCHOR 신생혈관생성 억제제(angiostatic agent); 엔도스타틴; De1-1 신생혈관형성 단백질; 씨티 3577; 콘토르트로스타틴(contortrostatin); 씨엠 101 ; 콘드로이티나아제 에이씨(chondroitinase AC); 씨디피 845; 칸스타틴; 비에스티 2002; 비에스티 2001; 비엘에스 0597; 비아이비에프 1000; 아레스틴(ARRESTIN); 아포미그렌(apomigren) (1304-1388-형 XV 콜라겐 (인간 유전자 COL15A1 알파l-쇄 전구체)); 안지오인히빈; aaATIII; 에이 36; 9알파-플루오로메드록시프로게스테론 아세테이트 ((6-알파)-17-(아세틸옥시)-9-플루오로-6-메틸-프레그-4-넨-3,20-디온); 2-메틸-2-프탈이미도-글루타르산 (2-(l,3-디히드로-l-옥소-2H-이소인돌-2-일)-2-메틸펜탄디온산); 이트륨 90 표지된 모노클로날 항체 비씨-1(Yttrium 90 labelled monoclonal antibody BC-1); 세막사니브(Semaxanib) (3-(4,5-디메틸피롤-2-일메틸렌)인돌린-2-온)(C15 H14 N2 O); 피아이 88 (포스포만노펜타오스 설페이트); 알보시디브(Alvocidib) (4H-1-벤조피란-4-온, 2-(2-클로로페닐)-5,7-디하이드록시-8-(3-하이드록시-l-메틸-4-피페리디닐)-시스-(-)-) (C21-H20 Cl N 05); 이 7820; 에스유 11248 (5-[3-플루오로-2-옥소-l,2-디하이드로인돌-(3Z)-일리덴메틸]-2,4-디메틸-lH-피롤-3-카르복실산 (2-디에틸아미노에틸)아미드) (C22 H27 F N4 O2); 스쿠알라민 (콜레스탄-7,24-디올, 3-[[3-[(4-아미노부틸)아미노프로필]아미노]-24-(하이드로겐 설페이트), (3.베타.,5.알파.,7.알파.)-) (C34 H65 N3 O5 S); 에리오크롬 블랙 티(Eriochrome Black T); 에이지엠 1470; (카르밤산, (클로로아세틸)-5-메톡시-4-[2-메틸-3-(3-메틸-2-부테닐)옥시라닐]-l-옥사피로[2,5]옥트-6-일 에스터, [3R-[3알파, 4알파(2R, 3R), 5베타, 6베타]]) (C 19 H28 Cl N 06); 에이지디 9935; 비아이비에프 1000; 에이지디 2171; 에이비티 828; 케이에스-인터류킨-2; 자궁글로빈(Uteroglobin); 에이 6; 엔에스씨 639366 (l-[3-(디에틸아미노)-2-하이드록시프로필아미노]-4-(옥시란-2-일메틸아미노)안트라퀴논 푸머레이트) (C24 H29 N3 O4. C4 H4 04); 아이에스브이 616; 항-ED-B 융합 단백질들; 에이취유아이 77; 트로포닌 I; 비씨-1 모노클로날 항체; 에스피브이 5.2; 이알 68203; 씨케이디 731 (3-(3,4,5-트리메톡시페닐)-2(E)-프로페논산 (3R,4S,5S,6R)-4-[2(R)-메틸-3(R)-3(R)-(3-메틸-2-부테닐)옥시란-2-일]-5-메톡시-l-옥사피로[2.5]옥트-6-일 에스터) (C28 H38 O8); 아이엠씨-1씨11(IMC-1Cl1; aaATIII; 에스씨 7; 씨엠 101; 안지오콜; 크링글 5(Kringle 5); 씨케이디 732 (3-[4-[2-(디메틸아미노)에톡시]페닐]-2(E)-프로페논산)(C29 H41 N 06); 유 995; 칸스타틴; 에스큐 885; 씨티 2584 (l-[l]-(도데실아미노)-10-하이드록시운데실]-3,7-디메틸크산틴)(C30 H55 N5 O3); 살모신(Salmosin); 이엠에이피 II; 티엑스 1920 (l-(4-메틸피페라지노)-2-(2-니트로-lH-l-이미다조일)-l-에탄온) (ClO Hl 5 N5 03); 알파-v 베타-x 저해제; 씨에이취이알 11509 (N-(1-프로피닐)글리실-[N-(2-나프틸)]글리실-[N-(카르바모일메틸)]글리신 비스(4-메톡시페닐)메틸아미드)(C36 H37 N5 O6); 비에스티 2002; 비에스티 2001; 비 0829; 에프알 111142; 4,5-디하이드록시-2(E)-헥사논산 (3R,4S,5S,6R)-4-[l(R),2(R)-에폭시-l,5-디메틸-4-헥세닐]-5-메톡시-l-옥사스피로[2.5]옥탄-6-일 에스터 (C22 H34 O7); 및 N-(4-클로로페닐)-4-(4-피리디닐메틸)-l-프탈아진아민; 4-[4-[[[[4-클로로-3-(트리플루오로메틸)페닐]아미노]카르보닐]아미노]페녹시]-N-메틸-2-피리딘카르복사미드; N-[2-(디에틸아미노)에틸]-5-[(5-플루오로-1,2-디하이드로-2-옥소-3H-인돌-3-일리덴)메틸]-2,4-디메틸-1H-피롤-3-카르복사미드; 3-[(4-브로모-2,6-디플루오로페닐)메톡시]-5-[[[[4-(1-피롤리디닐)부틸]아미노]카르보닐]아미노]-4-이소티아졸카르복사미드; N-(4-브로모-2-플루오로페닐)-6-메톡시-7-[(1-메틸-4-피페리디닐)메톡시]-4-퀴나졸린아민; 3-[5,6,7,13-테트라하이드로-9-[(1-메틸에톡시)메틸]-5-옥소-12H-인데노[2, 1-a]피롤로[3,4-c]카바졸-l 2-일]프로필 에스터 N,N-디메틸-글리신; N-[5-[[[5-(l,l-디메틸에틸)-2-옥사졸릴]메틸]티오]-2-티아졸릴]-4-피페리딘카르복사미드; N-[3-클로로-4-[(3-플루오로페닐)메톡시]페닐]-6-[5-[[[2-(메틸설포닐)에틸]아미노]메틸]-2-푸라닐]4-퀴나졸린아민; 4-[(4-메틸-피페라지닐)메틸]-N-[4-메틸-3-[[4-(3-피리디닐)-2-피리미디닐]아미노]-페닐]벤즈아미드; N-(3-클로로-4-플루오로페닐)-7-메톡시-6-[3-(4-몰포리닐)프로폭시]-4-퀴나졸린아민; N-(3-에티닐페닐)-6,7-비스(2-메톡시에톡시)-4-퀴나졸린아민; N-(3-((((2R)-l-메틸-2-피롤리디닐)메틸)옥시)-5-(트리플루오로메틸)페닐)-2-((3-(1,3-옥사졸-5-일)페닐)아미노)-3-피리딘카르복사미드; 2-(((4-플루오로페닐)메틸)아미노)-N-(3-((((2R)-l-메틸-2-피롤리디닐)메틸)옥시)-5-(트리플루오로메틸)페닐)-3-피리딘카르복사미드; N-[3-(아제티딘-3-일메톡시)-5-트리플루오로메틸-페닐]-2-(4-플루오로-벤질아미노)-니코틴아미드; 6-플루오로-N-(4-(1-메틸에틸)페닐)-2-((4-피리디닐메틸)아미노)-3-피리딘카르복사미드; 2-((4-피리디닐메틸)아미노)-N-(3-(((2S-)-2-피롤리디닐메틸)옥시)-5-(트리플루오로메틸)페닐)-3-피리딘카르복사미드; N-(3-(1,1-디메틸에틸)-lH-피라졸-5-일)-2-((4-피리디닐메틸)아미노)-3-피리딘카르복사미드; N-(3,3-디메틸-2,3-디하이드로-1-벤조퓨란-6-일)-2-((4-피리디닐메틸)아미노)-3-피리딘카르복사미드; N-(3-((((2S)-l-메틸-2-피롤리디닐)메틸)옥시)-5-(트리플루오로메틸)페닐)-2-((4-피리디닐메틸)아미노)-3-피리딘카르복사미드; 2-((4-피리디닐메틸)아미노)-N-(3-((2-(1-피롤리디닐)에틸)옥시)-4-(트리플루오로메틸)페닐)-3-피리딘카르복사미드; N-(3,3-디메틸-2,3-디하이드로-lH-인돌-6-일)-2-((4-피리디닐메틸)아미노)-3-피리딘카르복사미드; N-(4-(펜타플루오로에틸)-3-(((2S)-2-피롤리디닐메틸)옥시)페닐)-2-((4-피리디닐메틸)아미노)-3-피리딘카르복사미드; N-(3-((3-아제티디닐메틸)옥시)-5-(트리플루오로메틸)페닐)-2-((4-피리디닐-메틸)아미노)-3-피리딘카르복사미드; N-(3-(4-피페리디닐옥시)-5-(트리플루오로메틸)페닐)-2-((2-(3-피리디닐)에틸)아미노)-3-피리딘카르복사미드; N-(4,4-디메틸-l,2,3,4-테트라하이드로이소퀴놀린-7-일)-2-(lH-인다졸-6-일아미노)-니코틴아미드; 2-(1H-인다졸-6-일아미노)-N-[3-(1-메틸피롤리딘-2-일메톡시)-5-트리플루오로메틸-페닐]-니코틴아미드; N-[l-(2-디메틸아미노-아세틸)-3,3-디메틸-2,3-디하이드로-lH-인돌-6-일]-2-(lH-인다졸-6-일아미노)-니코틴아미드; 2-(1H-인다졸-6-일아미노)-N-[3-(피롤리딘-2-일메톡시)-5-트리플루오로메틸페닐]-니코틴아미드; N-(1-아세틸-3,3-디메틸-2,3-디하이드로-lH-인돌-6-일)-2-(lH-인다졸-6-일아미노)-니코틴아미드; N-(4,4-디메틸-l-옥소-1,2,3,4-테트라하이드로-이소퀴놀린-7-일)-2-(1H-인다졸-6-일아미노)-니코틴아미드; N-[4-(3차-부틸)-3-(3-피페리딜프로필)페닐][2-(1H-인다졸-6-일아미노)(3-피리딜)]카르복사미드; N-[5-(3차-부틸)이속사졸-3-일][2-(lH-인다졸-6-일아미노)(3-피리딜)]카르복사미드; 및 N-[4-(3차-부틸)페닐][2-(1H-인다졸-6-일아미노)(3-피리딜)]카르복사미드들을 포함하나, 이들에 제한되지 않는 키나아제 저해제들; 미합중국 특허 제6,258,812호; 동 제6,235,764호; 동 제6,630,500호; 동 제6,515,004호; 동 제6,713,485호; 동 제5,521,184호; 동 제5,770,599호; 동 제5,747,498호; 동 제5,990,141호; 미합중국 공개특허공보 제20030105091호; 및 국제특허협력조약 특허공개 제WO 01/37820호; 동 제WO 01/32651호; 동 제WO 02/68406호; 동 제WO 02/66470호; 동 제WO 02/55501호; 동 제WO 04/05279호; 동 제WO 04/07481호; 동 제WO 04/07458호; 동 제WO 04/09784호; 동 제WO 02/59110호; 동 제WO 99/45009호; 동 제WO 98/35958호; 동 제WO 00/59509호; 동 제WO 99/61422호; 동 제WO 00/12089호; 및 동 제WO 00/02871호들에 기술된 키나아제 저해제들이 포함되나, 이들에 제한되는 것은 아니며, 상기 공보들 각각은 전 목적들에 대하여 여기에 참조로 인용된다.
본 명세서에서 제공되는 바와 같은 항원 결합 단백질은 또한 성장 인자 저해제와 조합하여 사용될 수 있다. 이러한 약품들의 예들에는 EGF-R 항체들, EGF 항체들 및 EGF-R 저해제들인 분자들 등과 같은 EGF-R(상피세포 성장 인자 수용체)을 저해할 수 있는 약품들; VEGF 수용체들 및 VEGF를 저해할 수 있는 분자들 등과 같은 VEGF(혈관내피 성장 인자) 저해제들; 및 erbB2 수용체와 결합하는 유기분자들 또는 항체들, 예를 들면, 허셉틴™(HERCEPTIN™)(제넨테크 인코포레이티드) 등과 같은 erbB2 수용체 저해제들이 포함되나, 이들에 제한되는 것은 아니다. EGF-R 저해제들은, 예를 들면, 미합중국 특허 제5,747,498호, 국제특허공개공보 제WO 98/14451호, 동 제WO 95/19970호 및 동 제WO 98/02434호들에서 기술된 것들이다.
조합 요법(combination therapies)들의 특정의 예들에는, 예를 들면, 유방암 치료를 위한 탁솔 또는 탁산(예를 들면, 도세탁셀 또는 탁소테레) 또는 변형된 파클리탁셀(예를 들면, 아브락산 또는 오팍시오), 독소루비신 및/또는 아바스틴®과 함께의 c-fms 항원 결합 단백질; 신장암의 치료를 위한 다중표적항암제, 엠케이엘(수텐트, 넥사바르 또는 706) 및/또는 독소루비신과 함께의 인간 c-fms 항원 결합 단백질; 편평상피세포 암종의 치료를 위한 시스플라틴 및/또는 방사선과 함께의 c-fms 항원 결합 단백질; 폐암의 치료를 위한 탁솔 및/또는 카보플라틴과 함께의 c-fms 항원 결합 단백질들이 포함된다.
종양학에서의 적용예들에 더하여, 본 명세서에서 제공되는 상기 결합 단백질들은 염증성 질병들의 치료 또는 검출에서 사용될 수 있다. 마크로파지가 질병의 병리학에 기여하는 염증성 질병들에 있어서, 다른 세포상 격실(cellular compartments)들 내에서의 마크로파지들의 수준들을 감소시키는 상기 c-fms 항원 결합 단백질의 능력은 이들 질병들을 치료함에 있어서의 유용한 역할을 나타낸다. 인간 c-fms 항원 결합 단백질이 염증성 질병들, 예를 들면, 염증성 관절염(inflammatory arthritis), 죽상동맥경화증(atherosclerosis) 및 다발성 경화증(multiple sclerosis) 등에서의 조절에 있어서 어떤 역할을 한다는 것을 많은 연구들이 제안하고 있다.
치료될 수 있는 다른 질병들에는 염증성 장질활(inflammatory bowel disease), 크론씨병(Crohn's disease), 궤양성 대장염(ulcerative colitis), 류마티스성 척추염(rheumatoid spondylitis), 강직성 척추염(ankylosing spondylitis), 관절염(arthritis), 건선성 관절염(psoriatic arthritis), 류마티스성 관절염(rheumatoid arthritis), 퇴행성 관절염(osteoarthritis), 습진(eczema), 접촉성 피부염(contact dermatitis), 건선(psoriasis), 독성쇼크증후군(toxic shock syndrome), 패혈증(sepsis), 패혈성 쇼크(septic shock), 내독소 쇼크(endotoxic shock), 천식(asthma), 만성적 폐염증 질병(chronic pulmonary inflammatory disease), 규폐증(silicosis), 폐유육종(pulmonary sarcoidosis), 골다공증(osteoporosis), 재협착증(restenosis), 심장 및 신장의 재관류 손상(cardiac and renal reperfusion injury), 혈전증(thrombosis), 사구체신염(glomerulonephritis), 당뇨병(diabetes), 이식거부반응(graft vs. host reaction), 동종이식 거부반응(allograft rejection), 다발성 경화증(multiple sclerosis), 근변성(muscle degeneration), 근육퇴행위축(muscular dystrophy), 알쯔하이머병(Alzheimer's disease) 및 뇌졸증(stroke)들이 포함되나, 이들에 제한되는 것은 아니다.
마크로파지들에 의해 상산된 염증전 사이토카인(proinflammatory cytokines)들이 악액질 병리학에 포함될 것으로 여겨지기 때문에 상기 항원 결합 단백질들은 또한 악액질을 치료하는 데 사용될 수 있다(스위트(Sweet)와 그의 동료들의 2002, J. Immunol. 168:392-399; 보대르트(Boddaert)와 그의 동료들의 2006, Curr. Opin. Oncol. 8:335- 340 및 왕(Wang)과 그의 동료들의 2006 J. Endocrinology 190:415-423).
상기 항원 결합 단백질들의 능력으로 치료하는 데 사용되어야 할 여러 염증성 질병들이 주어지는 경우, 이들은 여러 다른 항염증제(anti-inflammatory agents)들과 함께 또는 이들과 결합되어 사용될 수 있다. 이러한 항염증제의 예들에는 TNF 약물들(예를 들면, 휴미라™(HUMIRA™), 레미케이드™(REMICADE™) 및 TNF 수용체 면역글로블린 분자들(엔브렐™(ENBREL™) 등과 같은) 등과 같은 TNF-알파 저해제들, IL-I 저해제들, 수용체 길항제들 또는 가용성 IL-lra(예를 들면, 키네레트(Kineret) 또는 아이씨이 저해제들(ICE inhibitors)), 앞서 기술된 바와 같은 콕스-2 저해제들 및 메탈로프로테아제 저해제들(COX-2 inhibitors and metalloprotease inhibitors) 및 알파-2-델타 리간드들(alpha-2-delta ligands)(예를 들면, 프레가발린™(PREGABALIN™) 및 뉴로틴™(NEUROTIN™))들이 포함되나, 이들에 제한되는 것은 아니다.
특정의 구체예들에 있어서, 항원 결합 단백질들은 또한 용골세포(osteoclast) 발달 및 활성화에서의 c-fms의 중요한 역할이라는 관점에서 여러 골질환들을 치료하는 데 사용될 수 있다(예를 들면, 롤프 에프(Rolf, F.)와 그의 동료들의 (2008) J. Biol. Chem. 55:340-349 및 와타른 에이(Watarn, A.)와 그의 동료들의 (2006) J. Bone Mineral Metabolism 24:274-282). 따라서, 상기 항원 결합 단백질들은 과다한 골 손실 또는 과도한 용골세포 활성이 요구되지 않는 경우에서 조차도 새로운 뼈의 형성을 필요로 하는 환자들을 포함하는 여러 의학적 장애들로 고통받는 환자들을 치료하는 데 유용할 수 있다. 과도한 용골세포 활성은 제공되는 상기 항원 결합 단백질들로 치료될 수 있는 다양한 골량 감소 장애들(osteopenic disorders)과 연관이 있으며, 이들 골량 감소 장애들에는 골량감소증(ostopenia), 골다공증, 치주염(periodontitis), 파제트씨병(Paget's disease), 부동태화로 인한 골 손실(bone loss due to immobilization), 골용해 전이(lytic bone metastases) 및 류마티스성 관절염, 강직성 척추염 및 뼈의 침식을 포함하는 다른 상태들을 포함하는 관절염들이 포함된다. 유방암 및 전립선암 등과 같은 일부 암들이 용골세포 활성을 증가시키고 그리고 뼈의 융식(bone resorption)을 유도하는 것으로 알려져 있다. 골수 내에서 야기되는 다발성 골수종이 또한 골 손실과 연관된다.
암의 골 전이(bone metastases)에 관하여는, 본 명세서에서 제공되는 상기 항원 결합 단백질들의 사용을 통한 상기 CSF-1/c-fms 축의 저해가 작용의 다중의 메카니즘들을 통하여 치료적인 잇점이 될 수 있다. 이들은 TAMs에 의하여 생산된 매트릭스분해효소(matrix degrading enzymes)들의 손실을 통한 침입 및 전이의 저해, 용골세포의 수들 및 기능의 손실을 통한 골수 내에서의 종양세포 시딩(seeding)에 대한 간섭, 앞서 언급된 TAMs들의 감소를 통한 전이 종양 성장의 저해 및 골 전이 영역들과 연관된 골 용해의 저해들이 포함될 수 있다(오노 에이취(Ohno, H.)와 그의 동료들의 (2008) Molecular Cancer Therapeutics. 5:2634-2643). 상기 항원 결합 단백질들은 또한 뼈의 암인 골육종(osteosarcoma)에 대한 치료적인 잇점들을 가질 수 있다.
당질코르티코이드 유도 골다공증, 이식 후 유도된 골다공증, 화학요법과 연관된 골다공증(즉, 화학요법 유도 골다공증), 부동태화 유도 골다공증, 기계적인 언로딩(mechanical unloading)으로 인한 골다공증 및 항경련제 사용과 연관되는 골다공증들을 포함하여 골다공증의 여러 형태들을 포함하여 다른 여러 낮은 골량 상태들이 또한 치료될 수 있다. 치료될 수 있는 다른 골 질병들에는 신부전과 연관되는 골 질병 및 영양, 위장 및/또는 간 연관의 골 질병들이 포함된다.
예를 들면, 퇴행성 관절염 및 류마티스성 관절염을 포함하여 관절염의 다른 형태들이 또한 치료될 수 있다. 상기 항원 결합 단백질들은 또한 관절염(예를 들면, 류마티스성 관절염)과 연관되는 전신적 골 손실을 치료하는 데 사용될 수 있다. 관절염의 치료에 있어서, 환자들은 상기 대상체 항원 결합 단백질들의 병변주변(perilesional) 또는 병변내(intralesional) 주사들에 의한 잇점을 가질 수 있다. 예를 들면, 상기 항원 결합 단백질은 염증화된 관절(inflamed joint)에 인접하거나 또는 그에 직접적으로 주사될 수 있으며, 따라서 그 사이트에서 손상된 뼈의 수복을 자극할 수 있다.
본 명세서에서 기술된 상기 항원 결합 단백질들은 또한 여러 뼈 수복 응용례들에서 사용될 수 있다. 예를 들면, 이들은 인공의 관절들과 연관된 마모편 골용해(wear debris osteolysis)를 지연시키고, 뼈 골절의 수복을 촉진하고, 그리고 골이식물(bone grafts)들이 그 안으로 이식되는 주변의 살아 있는 뼈들 내로의 골이식물들의 내포를 증강시키는 데 유용할 수 있다.
본 명세서에서 제공되는 상기 항원 결합 단백질들은, 골 장애들을 치료하는 데 사용되는 경우, 단독으로 또는 다른 치료제들과 함께, 예를 들면, 용골세포 활성을 억제하는 약품들을 갖거나 또는 용골세포 활성을 증가시키는 다른 약품들을 갖는 암요법제들과 함께 사용될 수 있다. 예를 들면, 상기 항원 결합 단백질들은 방사선요법 또는 화학요법을 수행하는 암환자들에게 투여될 수 있다. 상기 항원 결합 단백질들과 조합하여 사용되는 화학요법제들에는 안트라사이클린(anthracyclines), 탁솔, 타목시펜, 독소루비신, 5-플루오로우라실, 옥살로플라틴(oxaloplatin), 벨케이드® ([(lR)-3-메틸-l-[[(2S)-l-옥소-3-페닐-2-[(피라지닐카르보닐)아미노]프로필]아미노]부틸]보론산) 및/또는 암을 치료하는 데 사용되는 다른 작은 분자 약물들이 포함될 수 있다.
상기 항원 결합 단백질들은 골량의 손실의 결과를 가져오는 앞서 언급된 상태들의 치료에 대하여 단독으로 또는 BMP-1 내지 BMP-12로 표기되는 골 형태형성 인자들(bone morphogenic factors); 형질전환 성장 인자-베타(transforming growth factor-β) 및 TGF-β 구성원들; 섬유아세포 성장 인자들 FGF-1 내지 FGF-10; 인터루킨-1 저해제들(IL-1ra, antibodies to IL-1에 대한 항체들 및 IL-1 수용체들에 대한 항체들을 포함); TNFα 저해제들(에타네르셉트, 아달리부마브 및 인플릭시마브들을 포함); RANK 리간드 저해제들(가용성 RANK, 오스테오프로테게린 및 RANK 또는 RANK 리간드에 특이적으로 결합하는 길항적 항체들을 포함), Dkk-1 저해제들(예를 들면, 항-Dkk-1 항체들) 파라티로이드 호르몬, E 시리즈 프로스타글란딘, 비스포스포네이트 및 불소 및 칼슘 등과 같은 골-증강 미네랄들을 포함하나 이들에 제한되지 않는 골성장증강제(동화제) 또는 골 항-융식제(bone anti-resorptive agent)의 치료적 유효량과 조합으로 사용될 수 있다. 상기 항원 결합 단백질들 및 이들의 기능적인 단편들과 조합으로 사용될 수 있는 동화제(anabolic agents)들에는 파라티로이드 호르몬 및 인슐린-형 성장 인자(insulin-like growth factor ; IGF)들이 포함되며, 여기에서 후자의 약품들은 바람직하게는 IGF 결합 단백질과 착체로 된다. 이러한 조합 치료에 적절한 IL-1 수용체 길항제는 국제특허공개공보 제WO89/11540호에 기술되어 있으며, 적절한 가용성 TNF 수용체-1은 국제특허공개공보 제WO98/01555호에 기술되어 있다. 예시적인 RANK 리간드 길항제들은, 예를 들면, 국제특허공개공보 제03/086289호, 동 제WO 03/002713호, 미합중국 특허 제6,740,511호 및 동 제6,479,635호들에 기술되어 있다. 앞서 언급된 특허들 및 공개특허공보들 모두를 여기에 참조로 인용한다.
상기 항원 결합 단백질들은 또한 신생혈관생성(예를 들면, 종양 내)을 저해하는 데 사용될 수 있다. 예를 들면, 염증성 신생혈관생성이 1차적으로 FGF-2에 의해 작동되는 경우에서 상기 항원 결합 단백질들은 혈관 형성을 감소시키는 데 사용될 수 있다. 일부 구체예들에 있어서, 상기 항원 결합 단백질들은 VEGF 수준들이 낮고 그리고 종양 맥관 밀도가 높은 종양들 내에서의 신생혈관생성을 저해하는 데 사용된다.
진단적 방법들
기술된 상기 항원 결합 단백질들은 c-fms와 연관되는 질병들 및/또는 상태들을 검출하거나 진단하거나 또는 모니터링하기 위한 진단적인 목적들에 대하여 사용될 수 있다. 당해 기술분야에서 숙련된 자에게는 공지된 전통적인 면역조직학적 방법들을 사용하여 샘플 내에서의 c-fms의 존재의 검출에 대하여 기술된다(예를 들면, 티즈센의 1993, Practice and Theory of Enzyme Immunoassays, Vol 15(Eds R.H. Burdon and P.H. van Knippenberg, Elsevier, Amsterdam); 졸라(Zola)의 1987, Monoclonal Antibodies: A Manual of Techniques, pp. 147-158 (CRC Press, Inc.); 잘카넨(Jalkanen)과 그의 동료들의 1985, J. Cell. Biol. 101:976-985; 잘카넨과 그의 동료들의 1987, J. Cell Biol. 105:3087-3096). c-fms의 검출은 생체 내 또는 시험관 내에서 수행될 수 있다.
c-fms의 발현 및 상기 리간드들의 c-fms에의 결합을 검출하기 위한 상기 항원 결합 단백질들의 사용을 포함하는 진단적 응용례들이 본 명세서에서 제공된다. c-fms의 존재의 검출에서 유용한 방법들의 예들에는 효소면역측정법(ELISA ; enzyme linked immunosorbent assay) 및 방사면역측정법(RIA ; radioimmunoassay) 등과 같은 면역분석법(immunoassays)들이 포함된다.
진단적인 응용례들에 대하여, 상기 항원 결합 단백질은 전형적으로 검출가능한 표지기로 표지화될 것이다. 적절한 표지기들에는 방사성동위원소들 또는 방사성핵종들(예를 들면, 3H, 14C, 15N, 35S, 90Y, 99Tc, 111In, 125I, 131I), 형광기들(예를 들면, FITC, 로다민, 란탄계열 인광물질), 효소기들(예를 들면, 고추냉이과산화효소, 베타-갈락토시다아제, 루시퍼라아제, 알칼리포스파타아제), 화학발광기들, 비오티닐기들 또는 2차 리포터(예를 들면, 루신 지퍼 쌍 시퀀스들, 2차 항체들에 대한 바인딩 사이트들, 금속 바인딩 도메인들, 에피토프 태그들)에 의해 인식되는 사전설정된 폴리펩티드 에피토프들이 포함되나, 이들에 제한되는 것은 아니다. 일부 구체예들에 있어서, 상기 표지기는 잠재적인 입체장애를 감소시키기 위한 여러 길이들의 스페이서 아암들을 통하여 상기 항원 결합 단백질에 결합될 수 있다. 단백질들을 표지화하기 위한 여러 방법들이 당해 기술분야에서 공지되어 있으며, 사용될 수 있다.
다른 관점에 있어서, 항원 결합 단백질은 c-fms를 발현하는 세포 또는 세포들을 동정하는 데 사용될 수 있다. 특정의 구체예에 있어서, 상기 항원 결합 단백질은 표지기로 표지화되고, 그리고 상기 표지된 항원 결합 단백질의 c-fms에의 결합이 검출된다. 또 다른 특정의 구체예에 있어서, 상기 항원 결합 단백질의 c-fms에의 결합은 생체 내에서 검출된다. 또 다른 특정의 구체예에 있어서, 상기 c-fms 항원 결합 단백질은 단리되고 그리고 당해 기술분야에서 공지된 기술들을 사용하여 측정된다. 예를 들면, 하로우와 레인(Harlow and Lane)의 1988, Antibodies: A Laboratory Manual, New York: Cold Spring Harbor (ed. 1991 and periodic supplements); 존 이. 콜리간 저(John E. Coligan, ed.)의 1993, Current Protocols In Immunology New York: John Wiley & Sons)들을 참조하시오.
기술된 또 다른 관점은 제공된 상기 항원 결합 단백질들과 c-fms에의 결합에 대해 경쟁하는 시험 분자의 존재를 검출하기 위한 방법들을 기술한다. 이러한 분석의 하나의 구체예는 상기 시험 분자의 존재 또는 부재 중에서의 일정한 양의 c-fms를 포함하는 용액 내의 유리 항원 결합 단백질의 양을 검출하는 것을 포함한다. 유리 항원 결합 단백질(즉, c-fms에 부착되지 않은 상기 항원 결합 단백질)의 양의 증가는 상기 시험 분자가 c-fms에 대하여 상기 항원 결합 단백질과 경쟁할 수 있다는 것을 나타낼 수 있다. 하나의 구체예에 있어서, 상기 항원 결합 단백질은 표지기로 표지화된다. 달리, 상기 시험 분자가 표지화되고 그리고 항원 결합 단백질의 존재 및 부재 중에서 유리 시험 분자의 양이 모니터링된다.
치료의 방법들: 약제학적 제형들, 투여의 경로들
상기 항원 결합 단백질들을 사용하는 방법들이 또한 제공된다. 일부 방법들에 있어서, 항원 결합 단백질이 환자에게 제공된다. 상기 항원 결합 단백질은 인간 c-fms에 대한 CSF-1의 결합을 저해한다. 일부 방법들에 있어서의 항원 결합 단백질의 투여는 또한 CSF-1의 인간 c-fms에의 결합을 저해하는 것에 의하여 인간 c-fms의 자가인산화를 저해할 수 있다. 또한, 특정의 방법들에 있어서, 유효량의 적어도 하나의 항원 결합 단백질을 환자에게 투여하는 것에 의하여 단핵구 주화성이 감소된다. 일부 방법들에 있어서의 종양들 내로의 단핵구 이동이 유효량의 항원 결합 단백질의 투여에 의하여 저해된다. 게다가, 종양 또는 병이 든 조직 내에서의 종양 연관 마크로파지의 축적이 본 명세서에서 제공되는 바와 같은 항원 결합 단백질의 투여에 의하여 저해될 수 있다.
치료적 유효량의 하나 또는 복수의 상기 항원 결합 단백질들 및 약제학적으로 수용가능한 희석제(diluent), 담체(carrier), 가용화제(solubilizer), 에멀젼화제(emulsifier), 방부제(preservative) 및/또는 부형제(adjuvant)들을 포함하는 약제학적 조성물들이 또한 제공된다. 게다가, 이러한 약제학적 조성물을 투여하는 것에 의하여 환자를 치료하는 방법이 포함된다. 용어 "환자"는 인간 환자들을 포함한다.
수용가능한 제형물질(formulation materials)들은 사용된 투여량(dosages)들 및 농도들에서 공급받는 자(recipients)들에게 비독성이다. 특정의 구체예들에 있어서, 치료적 유효량의 인간 c-fms 항원 결합 단백질들을 포함하는 약제학적 조성물들이 제공된다.
특정의 구체예들에 있어서, 수용가능한 제형물질들은 바람직하게는 사용된 투여량 및 농도들에서 공급받는 자에게 비독성이다. 특정의 구체예들에 있어서, 상기 약제학적 조성물은, 예를 들면, 상기 조성물의 pH, 삼투압농도(osmolarity), 점도, 투명도, 색상, 등장도(isotonicity), 냄새, 멸균도(sterility), 안정성, 분해 또는 방출속도, 흡수 또는 투과도를 변형, 유지 또는 보존하기 위한 제형물질들을 포함할 수 있다. 이러한 구체예들에 있어서, 적절한 제형물질들에는 아미노산(글리신, 글루타민, 아스파라긴, 아르기닌 또는 리신 등과 같은); 항균제; 항산화제(아스코르빈산, 아황산나트륨 또는 아황산수소나트륨 등과 같은); 완충제(붕산염, 중탄산염, 트리스-염산, 시트르산염, 인산염 또는 다른 유기산들 등과 같은); 증량제(bulking agents ; 만니톨 또는 글리신 등과 같은); 킬레이트화제(에틸렌디아민테트라아세트산(EDTA)); 착화제(complexing agents ; 카페인, 폴리비닐피롤리돈, 베타-사이클로덱스트린 또는 히드록시프로필-베타-사이클로덱스트린); 충진제(fillers); 단당류; 이당류; 및 다른 탄수화물(글루코스, 만노스 또는 덱스트린 등과 같은); 단백질(혈청알부민, 젤라틴 또는 면역글로블린 등과 같은); 착색제, 착향제 및 희석제; 에멀젼화제; 친수성 중합체(폴리비닐피롤리돈 등과 같은); 저분자량 폴리펩티드; 염-형성 짝이온(salt-forming counterions ; 나트륨 등과 같은); 방부제(벤잘코늄클로라이드, 안식향산, 살리실산, 티메로살, 페네틸알코올, 메틸파라벤, 프로필파라벤, 클로르헥시딘, 소르빈산 또는 과산화수소 등과 같은); 용제(글리세린, 프로필렌글리콜 또는 폴리에틸렌글리콜 등과 같은); 당알코올(만니톨 또는 소르비톨 등과 같은); 현탁화제; 계면활성제 또는 적심제(wetting agents)(플루로닉, 폴리에틸렌글리콜, 소르비탄에스테르, 폴리소르베이트 20 등과 같은 폴리소르베이트, 트리톤, 트로메타민, 레시틴, 콜레스테롤, 틸록사팔 등과 같은); 안정성증강제(슈크로스 또는 소르비톨 등과 같은); 장력증강제(tonicity enhancing agents ; 알칼리금속 할로겐화물, 바람직하게는 염화나트륨 또는 염화칼륨, 만니톨, 소르비톨 등과 같은); 전달비히클(delivery vehicles); 희석제; 첨가제(excipients) 및/또는 약제학적 부형제들이 포함되나, 이들에 제한되는 것은 아니다. 레밍턴의 약제학(REMINGTON'S PHARMACEUTICAL SCIENCES, 18" Edition, (A.R. Genrmo, ed.), 1990, Mack Publishing Company)을 참조하시오.
특정의 구체예들에 있어서, 적절한 약제학적 조성물은 당해 기술분야에 숙련된 자에 의하여, 예를 들면, 목적하는 투여의 경로, 전달방식(delivery format) 및 원하는 투여량에 따라 결정될 수 있을 것이다. 예를 들면, 위의 레밍턴의 약제학을 참조하시오. 특정의 구체예들에 있어서, 이러한 조성물들은 물리적인 상태, 안정성, 기술되는 상기 항원 결합 단백질들의 생체 내 방출 속도 및 생체 내 정화값(clearance)에 영향을 줄 수 있다. 특정의 구체예들에 있어서, 약제학적 조성물 내의 1차 비히클(primary vehicle) 또는 담체는 속성에 있어서 수성 또는 비-수성이 될 수 있다. 예를 들면, 적절한 비히클 또는 담체는 주사를 위한 물, 비경구투여(parenteral administration)를 위한 조성물들에서 공통적인 다른 물질들로 보충된 생리식염수 또는 인공뇌척수액(artificial cerebrospinal fluid)들이 될 수 있다. 중성의 완충된 식염수 또는 혈청알부민과 혼합된 식염수들이 다른 예시적인 비히클들이다. 특정의 구체예들에 있어서, 약제학적 조성물들은 대략 pH 7.0 내지 8.5의 트리스 완충제 또는 대략 pH 4.0 내지 5.5의 아세테이트 완충제를 포함하며, 또한 소르비톨 또는 적절한 대체물(substitute)을 더 포함할 수 있다. 특정의 구체예들에 있어서, 인간 c-fms 항원 결합 단백질 조성물들은 선택적인 제형화제(formulation agents)(위의 레밍턴의 약제학)와 함께 원하는 순도를 갖는 선택된 조성물을 동결건조된 케이크(lyophilized cake) 또는 수성용액의 형태로 혼합하는 것에 의하여 저장용으로 제조될 수 있다. 더욱이, 특정의 구체예들에 있어서, 상기 인간 c-fms 항원 결합 단백질은 슈크로스 등과 같은 적절한 첨가제들을 사용하여 동결건조물(lyopholizate)로서 제형화될 수 있다.
상기 약제학적 조성물들은 비경구적 전달용으로 선택될 수 있다. 달리, 상기 조성물들은 흡입용 또는 경구적으로 등과 같이 소화관(digestive tract)을 통한 전달용으로 선택될 수 있다. 이러한 약제학적으로 수용가능한 조성물들의 제조는 당해 기술분야에서의 기술범위 이내이다.
제형화 구성성분(formulation components)들은 바람직하게는 투여의 장소에 받아들여질 수 있는 농도들로 존재한다. 특정의 구체예들에 있어서, 완충제들이 사용되어 생리학적인 pH에서 또는 약간 낮은 pH에서, 전형적으로는 약 5 내지 약 8의 범위의 pH에서 상기 조성물이 유지되도록 한다.
비경구적 투여가 고려되는 경우, 치료적인 조성물(therapeutic compositions)들은 약제학적으로 수용가능한 비히클 내에 원하는 인간 c-fms 항원 결합 단백질을 포함하는 무-발열인자(pyrogen-free)의, 비경구적으로 수용가능한 수성용액의 형태로 제공될 수 있다. 비경구적 주사를 위한 특히 적절한 비히클은 멸균 증류수이며 그 안에 상기 인간 c-fms 항원 결합 단백질이 멸균, 등장의, 적절하게 방부처리된 용액으로서 제형화된다. 특정의 구체예들에 있어서, 조제용 물질(preparation)은 주사가능한 미소구(microspheres), 생분해성(bioerodible)의 입자, 중합체성 화합물(폴리유산 또는 폴리글리콜산 등과 같은), 비드(beads) 또는 리포좀 등과 같은 약품들과 함께 원하는 분자의 제형을 포함할 수 있으며, 이들은 데포주사(depot injection)을 통하여 전달될 수 있는 제품의 제어되거나 또는 지속되는 방출을 제공할 수 있다. 특정의 구체예들에 있어서, 히알루론산이 또한 사용되어 순환에 있어서 지속되는 내구기간(sustained duration)의 효과를 갖도록 할 수 있다. 특정의 구체예들에 있어서, 이식가능한 약물 전달 기구들이 사용되어 원하는 항원 결합 단백질을 도입하도록 할 수 있다.
특정의 약제학적 조성물들은 흡입용으로 제형화된다. 일부 구체예들에 있어서, 인간 c-fms 항원 결합 단백질들은 건조된, 흡입가능한 분말로 제형화된다. 특정의 구체예들에 있어서, 인간 c-fms 항원 결합 단백질 흡입 용액들이 또한 에어로졸 전달(aerosol delivery)을 위한 추진제와 함께 제형될 수 있다. 특정의 구체예들에 있어서, 용액들은 또한 분무될 수 있다. 따라서, 폐 투여 및 제형화 방법은 국제특허출원 제PCT/US94/001875호에 더욱 기술되며, 이는 여기에 참조로 인용되며, 또한 화학적으로 변형된 단백질들의 폐 전달을 기술하고 있다. 일부 제형들은 경구적으로 투여될 수 있다. 이러한 방법으로 투여되는 인간 c-fms 항원 결합 단백질들은 정제 또는 캡슐 등과 같은 고형 투여량 형태(solid dosage forms)의 컴파운딩(compoundig)에사 상용적으로 사용되는 담체들과 함께 또는 담체들 없이 제형화될 수 있다. 특정의 구체예들에 있어서, 캡슐은 생물학적 이용가능성(bioavailability)이 극대화 되고 그리고 전전신적 분해(pre-systemic degradation)가 최소화될 때 위장관의 지점에서 제형의 상기 활성부분을 방출하도록 설계될 수 있다. 상기 인간 c-fms 항원 결합 단백질의 흡수를 용이하게 하기 위한 별도의 약제들이 포함될 수 있다. 희석제, 착향제, 저융점의 왁스, 식물성 오일, 윤활제, 현탁화제, 정제 붕해제 및 결합제들이 또한 사용될 수 있다.
일부 약제학적 조성물들은 정제들의 제조에 적절한 비독성의 부형제들과의 혼합물 내에 유효량의 하나 또는 복수의 인간 c-fms 항원 결합 단백질들을 포함한다. 멸균수 또는 다른 적절한 비히클 내에 상기 정제들을 용해시키는 것에 의하여, 용액들이 단위-투여량 형태(unit-dose form)로 제조될 수 있다. 적절한 부형제들에는 탄산칼슘, 탄산나트륨 또는 중탄산나트륨, 락토오스 또는 인산칼슘 등과 같은 비활성 희석제; 또는 전분, 젤라틴 또는 아카시아(acacia); 또는 스테아린산마그네슘, 스테아린산 또는 활석(talc) 등과 같은 윤활제들이 포함되나, 이들에 제한되는 것은 아니다.
인간 c-fms 항원 결합 단백질들을 포함하는, 지연-전달 또는 제어-전달 제형들로의 추가의 약제학적 조성물들은 당해 기술분야에서 숙련된 자들에게는 명백할 것이다. 리포좀 담체(liposome carriers), 생분해성 마이크로입자(bio-erodible microparticles) 또는 다공성 비드(porous beads) 및 데포 주사 등과 같은 다양한 다른 지연-전달 또는 제어-전달의 수단으로 제형화하는 기술들이 당해 기술분야에서 숙련된 자들에게는 또한 공지된 것이다. 예를 들면, 참조로 인용되며 또한 약제학적 조성물들의 전달을 위한 다공성의 중합체성 마이크로입자들의 제어된 방출을 기술하는 국제특허출원 제PCT/US93/00829호를 참조하시오. 지연-방출 제제들은 형상화된 물품들, 예를 들면, 필름 또는 마이크로캡슐의 형태의 반투과성의 중합체 매트릭스(semipermeable polymer matrices)를 포함할 수 있다. 지연 방출 매트릭스들에는 폴리에스터, 하이드로겔(hydrogels), 폴리락티드(각각 참조로 인용되는 미합중국 특허 제3,773,919호 및 유럽특허공개공보 제EP 058481호 등과 같은), L-글루탐산과 감마 에틸-L-글루타메이트의 공중합체(시드만(Sidman)과 그의 동료들의 1983, Biopolymers 2:547-556), 폴리(2-하이드록시에틸린에타크릴레이트)(랑거(Langer)와 그의 동료들의 1981, J. Biomed. Mater. Res. 15:167-277 및 랑거의 1982, Chem. Tech. 12:98-105), 에틸렌비닐아세테이트(랑거와 그의 동료들의 위의 1981 문헌) 또는 폴리-D(-)-3-하이드록시부틸산(유럽특허공개공보 제EP 133,988호)들이 포함될 수 있다. 지연 방출 조성물들에는 또한 당해 기술분야에서 공지된 여러 기술들 중의 임의의 것에 의해 제조될 수 있는 리포좀들이 포함될 수 있다. 예를 들면, 참조로 인용되는 엡스타인(Eppstein)과 그의 동료들의 1985, Proc. Natl. Acad. Sci. U.S.A. 82:3688-3692; 유럽특허공개공보 제EP 036,676호; 동 제EP 088,046호 및 동 제EP 143,949호들을 참조하시오.
생체 내 투여를 위하여 사용되는 약제학적 조성물들은 전형적으로는 멸균 제제들로서 제공된다. 멸균(sterilization)은 멸균여과막(sterile filtration membranes)을 통한 여과에 의해 수행될 수 있다. 상기 조성물이 동결건조되는 경우, 이러한 방법을 사용하는 멸균이 동결건조 및 재구성(reconstitution) 전 또는 후 둘 중 어느 하나에서 수행될 수 있다. 비경구투여를 위한 조성물들은 동결건조된 형태 또는 용액으로 저장될 수 있다. 비경구적 조성물들은 대체로 멸균 접근 구명(sterile access port)을 구비한 용기, 예를 들어 피하 주사바늘로 찔러 넣을 수 있는 스토퍼(stopper)를 구비한 바이알 또는 정맥내 용액백 내에 보관한다.
특정의 구체예들에 있어서, 본 명세서에서 기술되는 바와 같은 재조합 항원 결합 단백질을 발현하는 세포들은 전달을 위하여 캡슐화(encapsulated) 된다(Invest. Ophthalmol Vis Sci 43:3292-3298, 2002 및 Proc. Natl. Acad. Sciences 103:3896-3901, 2006들을 참조하시오).
특정의 제형들에 있어서, 항원 결합 단백질은 적어도 10㎎/㎖, 20㎎/㎖, 30㎎/㎖, 40㎎/㎖, 50㎎/㎖, 60㎎/㎖, 70㎎/㎖, 80㎎/㎖, 90㎎/㎖, 100㎎/㎖ 또는 150㎎/㎖의 농도를 갖는다. 일부 제형들은 완충제, 슈크로서 및 폴리소르베이트를 포함한다. 제형의 하나의 구체예는 50 내지 100㎎/㎖의 항원 결합 단백질, 5 내지 20mM의 소듐아세테이트, 5 내지 10중량/용적%의 슈크로스 및 0.002 내지 0.008중량/용적%의 폴리소르베이트를 포함하는 것이다. 예를 들면, 특정의 제형은 9 내지 11mM의 소듐아세테이트 완충제 내의 65 내지 75㎎/㎖의 항원 결합 단백질, 8 내지 10중량/용적%의 슈크로스 및 0.005 내지 0.006중량/용적%의 폴리소르베이트를 포함한다. 이러한 특정의 제형들의 pH는 4.5 내지 6의 범위 이내이다. 다른 제형들은 5.0 내지 5.5의 pH(예를 들면, 5.0, 5.2 또는 5.4의 pH)를 갖는다.
일단 상기 제약학적 조성물이 제형화되면, 이는 용액, 현탁액, 겔, 에멀젼, 고체, 결정으로서 또는 탈수되거나 또는 동결건조된 분말로서 멸균 바이알(sterile vials) 내에 저장될 수 있다. 이러한 제형들은 즉시사용형 형태(ready-to-use form)으로 또는 투여에 앞서 재구성되는 형태(예를 들면, 동결건조된 형태)로 저장될 수 있다. 1회-투여량 투여 유닛(single-dose administration unit)을 생산하기 위한 키트(kits)들이 또한 제공된다. 특정의 키트들은 건조된 단백질을 갖는 제1용기 및 수성 제형을 갖는 제2용기를 포함한다. 특정의 구체예들에 있어서, 단일 및 다중-격실의 사전-충진된 주사기들(예를 들면, 액체 주사기들 및 리오시린지(lyosyringes))을 포함하는 키트들이 제공된다. 사용될 인간 c-fms 항원 결합 단백질을 포함하는 약제학적 조성물의 상기 치료적 유효량은, 예를 들면, 치료적인 정황(therapeutic context) 및 목표(objectives)들에 의존적일 수 있다. 당해 기술분야에서 숙련된 자들에게는 치료를 위한 적절한 투여량 수준들이 부분적으로는 전달되는 상기 분자, 상기 인간 c-fms 항원 결합 단백질이 사용되는 징후, 투여의 경로 및 환자의 크기(체중, 체표면적 또는 장기 크기) 및/또는 환자의 상태(연령 및 일반적인 건강)에 의존적일 수 있다는 것은 이해될 수 있는 것이다. 특정의 구체예들에 있어서, 임상의는 적정한 치료 효과를 얻기 위하여 투여량을 적정하고 투여 경로를 변경할 수 있다.
전형적인 투여량은 앞서 언급한 상기 인자들에 따라 약 1㎍/㎏ 내지 약 30㎎/㎏의 범위가 될 수 있다. 특정의 구체예들에 있어서, 상기 투여량은 10㎍/㎏ 내지 30㎎/㎏, 적절하게는 0.1㎎/㎏ 내지 약 30㎎/㎏, 달리 0.3㎎/㎏ 내지 약 20㎎/㎏의 범위가 될 수 있다. 일부 적용예들에 있어서, 상기 투여량은 0.5㎎/㎏ 내지 20㎎/㎏이다. 일부 경우들에 있어서, 항원 결합 단백질은 0.3㎎/㎏, 0.5㎎/㎏, 1㎎/㎏, 3㎎/㎏, 10㎎/㎏ 또는 20㎎/㎏으로 투여된다. 일부 치료 체계(treatment regimes)들에서의 상기 투여량 스케쥴(dosage schedule)은 주당(qW) 0.3㎎/㎏, 주당 0.5㎎/㎏, 주당 1㎎/㎏, 주당 3㎎/㎏, 주당 10㎎/㎏ 또는 주당 20㎎/㎏의 투여량이다.
투여주기(dosing frequency)는 사용되는 제형 내의 특정한 인간 c-fms 항원 결합 단백질의 약물동력학적 변수(pharmacokinetic parameters)들에 의존적일 수 있다. 전형적으로, 임상의는 원하는 효과를 달성하는 투여량에 달할 때까지 상기 조성물을 투여한다. 따라서, 상기 조성물은 단일 투여량으로서, 또는 2회 또는 그 이상의 투여량들(이는 상기 원하는 분자의 동일량을 포함하거나 또는 포함하지 않을 수 있음)을 시간경과에 따라 또는 이식기구(implantation device) 또는 카테터를 통하여 연속적인 투입으로 투여될 수 있다. 적절한 투여량들은 적절한 투여-반응 데이터의 사용을 통하여 알아낼 수 있다. 특정의 구체예들에 있어서, 상기 항원 결합 단백질들은 연장된 시간의 기간을 통하여 환자들에게 투여될 수 있다. 항원 결합 단백질의 만성적 투여는 통상적으로 완전한 인간이 아닌, 예를 들면, 비-인간의 동물 내에서 인간 항원, 예를 들면, 불완전한 인간 항원 또는 비-인간의 종들 내에서 생산되는 비-인간의 항체인 항원 결합 단백질들과 연관되는 면역부작용 또는 알러지반응들을 최소화한다.
상기 약제학적 조성물의 투여의 경로는 공지의 방법들, 예를 들면, 경구적으로, 정맥내, 복강내, 뇌내(유조직내(intra- parenchymal)), 뇌실내(intracerebroventricular), 근내(intramuscular), 안내(intra-ocular), 동맥내, 문맥내(intraportal) 또는 병소내 주사를 통하여; 지연 방출 시스템들에 의하거나 또는 이식기구들에 의하는 것들에 따른다. 특정의 구체예들에 있어서, 상기 조성물들은 일시주사 또는 투입에 의하여 연속적으로 또는 이식기구에 의하여 투여될 수 있다.
상기 조성물은 또한 그 위에 원하는 분자가 흡수되거나 또는 캡슐화된 막, 스펀지 또는 다른 적절한 물질의 이식을 통하여 국소적으로 투여될 수 있다. 특정의 구체예들에 있어서, 이식기구가 사용되는 경우, 상기 기구는 임의의 적절한 조직 또는 장기 내로 이식될 수 있으며, 원하는 분자의 전달은 확산, 시간 지연된 일시투여 또는 연속적인 투여를 통할 수 있다.
기술된 생체 외에 따라 인간 c-fms 항원 결합 단백질 약제학적 조성물들을 사용하는 것이 또한 바람직할 수 있다. 이러한 경우들에 있어서, 환자로부터 제거되는 세포들, 조직들 또는 장기들을 인간 c-fms 항원 결합 단백질 약제학적 조성물들에 노출시킨 후, 그 세포들, 조직들 및/또는 장기들을 후속적으로 다시 환자에게 이식한다.
특히, 인간 c-fms 항원 결합 단백질들은 본 명세서에서 기술된 것들과 같은 방법들을 사용하여 상기 폴리펩티드를 발현하고 그리고 분비하는, 유전적으로 조작된 특정의 세포들을 이식하는 것에 의하여 전달될 수 있다. 특정의 구체예들에 있어서, 이러한 세포ㄷ르은 동물 또는 인간 세포들이 될 수 있으며, 또한 자가조직(autologous)의, 이종조직(heterologous)의 또는 이종발생(xenogeneic)의 것들이 될 수 있다. 특정의 구체예들에 있어서, 상기 세포들은 불멸화될 수 있다. 다른 구체예들에 있어서, 면역반응의 기회를 감소시키기 위하여, 상기 세포들을 캡슐화하여 주변의 조직들의 투과(infiltration)를 피하도록 할 수 있다. 다른 구체예들에 있어서, 상기 캡슐화 물질들은 전형적으로는 상기 단백질 생성물(들)의 방출은 허용하나 환자의 면역체계에 의하거나 또는 상기 주변 조직들로부터의 다른 해로운 인자들에 의한 파괴를 방지하는, 생체적합성의, 반투과성의 중합체성 인클로져(enclosures) 또는 막들이다.
수행된 실험들 및 달성된 결과들과 함께 하기의 실시예들은 단지 설명을 위한 목적들로 제공되는 것이며, 첨부된 특허청구범위들의 관점을 제한하는 것으로 의도되는 것은 아니다.
실시예
분석
AML-5 분석
항체들이 직접적으로 c-fms에 대하여 결합하고 그리고 c-fms/CSF-1 축을 차단함에 있어서의 기능적인 활성을 나타내는 지의 여부를 결정하기 위하여, 세포-기반의 생물검정(bioassay)을 사용하였다.
이 분석은 인간 골수단구성 세포주(human myelomonocytic cell line), AML5(미합중국 온타리오 토론토 소재 유니버시티 헬스 네트워크) 의존성 성장인자의 CSF-1-구동의 증식을 정량적으로 측정한다. 따라서, 상기 분석은 이 경로를 차단하는 약품들을 도입하는 것에 의한 이러한 증식의 억제를 측정한다. 이러한 분석에 있어서, AML-5 세포들을 감소하는 농도들의 항체의 존재 중에서 10ng/㎖ CSF-1과 함께 배양시켰다. 72시간 후, 상기 세포들의 대사 활성에 기초하는 증식의 간접적인 측정인 알라마르 블루™(Alamar Blue™ ; 바이오소스(Biosource))를 사용하여 세포 증식을 측정하였다.
골수 분석
상기 항체들이 사이노몰거스 원숭이 c-fms와 교차-반응하는 지의 여부를 결정하기 위한 유사한 분석에 있어서, 1차적인 원숭이 골수로부터의 단핵구성 세포들의 CSF-1-구동의 증식에서 항체들을 시험하였다. 상기 AML-5 증식 분석과 유사하게, 사이노몰거스 골수 세포들을 감소하는 농도들의 항체의 존재 중에서 10ng/㎖ CSF-1과 함께 배양시켰다. 72시간 후, 세포증식을 알라마르 블루를 사용하여 측정하였다.
실험들에서 사용된 항체 클론들
하기의 실험들은 1.109, 1.2, 2.360으로 표시되는 3개의 항체 클론들의 사용을 포함하며, 이들은 모두 2개의 완전한 중쇄 및 2개의 완전한 경쇄들을 포함하는 사량체들이다. 클론 1.109는 2개의 중쇄 H1(SEQ ID NO: 4) 및 2개의 경쇄 L1(SEQ ID NO: 36)들을 포함하고, 클론 1.2는 2개의 중쇄 H8(SEQ ID NO: 11) 및 2개의 경쇄 L8(SEQ ID NO: 43)들을 포함하고, 그리고 클론 2.360은 2개의 중쇄 H24(SEQ ID NO: 27) 및 2개의 경쇄 L22(SEQ ID NO: 57)들을 포함한다.
실시예
1 : c-
fms
하이브리도마들의
준비
구체예들은 인간 cfms에 대하여 지향되는 완전한 인간 모노클로날 항체들을 제조하기 위하여 제노마우스®(XenoMouse®) 기술을 사용할 수 있다. 면역화의 목적들을 위하여, C-말단 인간 Fc 도메인을 갖는 c-fms-Fc, 인간 c-fms 세포외 도메인(잔기 1-512, 도 8; SEQ ID: 1을 참조)들을 사용하였다. 게다가, c-fms-LZ, c-말단 류신 지퍼 도메인(암겐 롯트 번호 45640-43) 및 293ㅆ/c-fms를 갖는 인간 c-fms 세포외 도메인(잔기 1-512), 전장의 인간 c-fms로 감염된 인간 아데노바이러스 5형-형질전환된 인간 배아 신장세포주(human adenovirus type 5-transformed human embryonic kidney cell line)들이 상기 항-c-fms 항체들을 스크리닝 하는 데 활용되었다.
코호트 1(cohort 1 (IgG1) 및 코호트 2(IgG2) 제노마이스®들을 c-fms-Fc로 면역화/부스트(immunized/boosted)시켰다. 효소면역측정법(ELISA)에 의해 혈청 적정량(serum titers)들을 측정하였으며, 코호트 1 및 코호트 2 둘 다로부터의 비장들을 융합시켜 하이브리도마들을 생성시켰다. 그 결과의 폴리클로날 상청액들을 c-fms-LZ에의 결합에 대하여는 효소면역측정법에 의하여 그리고 293T/c-fms 세포들은 형광미세용적분석기술(FMAT ; fluorometric microvolume assay technology)에 의하여 스크리닝하였다. 형광-활성화세포분류(FACS ; fluorescence-activated cell sorting)에 의하여 c-fms/293T 세포들에의 CSF-1의 결합의 저해에 대하여 총 828개의 양성의 상청액들이 시험되었다. 그 결과의 168개의 양성의 상청액들이 급성 골수성 백혈병(AML ; acute myelogenous leukemia)-5 세포들의 CSF-1-유도 증식의 저해에 대하여 더 시험되었다. 스크리닝에 기초하여, 33개의 하이브리도마들이 CSF-1 활성에 길항적인 것으로 동정되었으며, 복제를 위하여 선택되었다.
실시예 2 : 항-c-
fms
하이브리도마들의
특정화
상기 33개의 선택된 하이브리도마들로부터, 29개(19개의 IgG1 및 10개의 IgG2 아이소타입들)들을 성공적으로 복재하였으며, 이들 클론들로부터의 상청액들을 상기 293T/c-fms에의 CSF-1 결합의 저해 및 AML-5 세포들의 CSF-1 유도 증식의 저해에 대하여 시험하였다. 단량체성 c-fms 단백질을 사용하는 저-해상도 바이어코어 결합 분석(low-resolution Biacore binding assay)은 이들 29개의 항-c-fms 하이브리도마들의 KD가 0.1 내지 43nM의 범위 이내에 있음을 나타내었다(표 8(항-c-fms 하이브리도마들에 대한 저 해상도 바이어코어 결합 분석 결과들)을 참조). 항-인간 IgG를 아민 커플링(amine coupling)을 사용하는 센서칩(sensor chip)의 4개의 플로우셀(flow cells)들 모두의 위에 부동태화시켰다. 조 하이브리도마 샘플들을 절반으로 희석시키고 그리고 상기 항-IgG 표면 상에 포착시켰다. 단량체성 c-fms(잔기 1-512)-pHis가 125nM의 농도에서의 검출대상물(analyte) 이었다. 시퀀스 계통 분석(Sequence lineage analyses)들이 또한 상기 29개의 하이브리도마들 상에서 수행되었다(표 2를 참조).
[표 8-1]
[표 8-2]
상기 결합 저해 및 증식 저해 분석들에 기초하여, 상기 29개의 상청액들 중의 16개(11개의 IgG1 및 5개의 IgG2 아이소타입들)를 더욱 특정화하기 위하여 선택하였다. 각각 생쥐 DRM(생쥐 골수의 덱스터 형 배양물(Dexter type culture)로부터 유도된 ras- 및 myc- 불멸화 단핵구성 세포주(monocytic cell line)) 세포들 및 1차의 사이노몰거스 골수세포들의 CSF-1 유도 증식의 억제에 의해 생쥐 및 사이노몰거스 c-fms에 대한 교차-반응성을 시험하였다. 세포 증식에 대하여는, 상기 상청액들 중 어느 것도 상기 생쥐 DRM 세포들의 증식을 억제하지 않은 반면에(데이터는 나타내지 않음), 16개의 상청액들 중 13개는 상기 사이노몰거스 골수 세포들의 증식을 억제하였다. 상기 상청액들을 또한 인간 말초혈액-유도 CD14+ 단핵구들의 CSF-1-유도 증식의 억제에 대해 시험하였으며, 인간 AML-5 생물검정에서 재시험하였으며(앞서의 분석 영역을 참조), 그 결과들을 표 9(상기 항-c-fms 하이브리도마 상청액들에 대한 생물검정 결과들의 요약) 및 표 10(항-c-fms 하이브리도마 상청액들에 대한 생물검정 결과들의 개요)에 나타내었다. 집쥐 항-인간 c-fms 항체인 2-4A5 항체(바이오소스(Biosource)))를 양성 대조(positive control)로 사용하였다.
4개의 IgG1 아이소타입 항체들이 상기 AML-5 생물검정에서 10pM 미만(<10pM)의 포텐시(potency)를 가졌으며, 상기 항체들 중 3개, 즉 클론 ID No. 1.2.1, 1.109.3 및 1.134.1들은 사이노몰거스 골수 세포들의 증식을 저해하였다. 상기 3개의 항체들 중, 클론 1.2.1 및 1.109.3들은 상기 바이어코어 결합 분석에서 c-fms에 대해 가장 높은 친화도를 가졌다. 2개의 IgG2 아이소타입 항체들인 2.103.3 및 2.360.2들은 상기 AML-5 및 사이노몰거스 골수 생물검정들에서 높은 포텐시를 나타내었으며, 상기 바이어코어 분석에서 c-fms에 대하여 유사한 친화도를 나타내었다. 상기 AML-5 및 사이노몰거스 골수 생물검정들에 있어서 높은 포텐시를 나타내는 상기 5개의 항체들은 또한 시퀀스에서 다양성을 나타냈다. 이들 포텐시, 친화도 및 다양성의 인자들에 기초하여, 클론 1.2.1, 1.109.3 및 2.360.2들이 추가의 전개 및 특정을 위하여 선택되었다.
[표 9]
[표 10]
실시예
3 : 항체들의 발현 및 특정화
항체 클론들 1.2, 1.109 및 2.360들에 대한 중쇄 및 경쇄 유전자들을 단리시키고 그리고 IgG2 중쇄들 및 카파 경쇄들로서의 발현을 위한 구축물들 내로 복제시켰다. COS/PKB 내에서의 일시적 발현에 의해 항체들을 발현시켰으며, 단백질 A 크로마토그래피(Protein A chromatography)로 정제하였다. 항체 수율들은 3.6 내지 7.4㎎/ℓ이었으며, 이는 이러한 발현 시스템에 대하여 기대되는 범위 이내이다.
상기 복제된 항체들 및 상기 하이브리도마-발현 항체들의 활성들을 AML-5 증식 분석(AML-5 proliferation assay)에서 비교하였다. AML-5 세포들을 감소하는 농도들의 항체의 존재 중에서 10ng/㎖ CSF-1과 함께 배양시켰다. 72시간 후, 알라마르 블루를 사용하여 세포 증식을 측정하였다(도 3 참조). 상기 재조합 항체들은 상기 하이브리도마 상청액들과 유사한 중화 활성(neutralizing activity)을 나타내었으며, IgG1 내지 IgG2들로부터의 1.2 및 1.109의 전환은 뚜렷한 효과를 갖지 못하였다. 상기 재조합 항체들은 또한 도 4에 나타낸 바와 같이 사이노몰거스 증식에서 양호한 중화 활성을 입증하였다. 상기 AML-5 증식 분석과 유사하게, 사이노몰거스 골수 세포들을 감소하는 농도들의 항체의 존재 중에서 10ng/㎖ CSF-1과 함께 배양시켰다. 72시간 후, 알라마르 블루를 사용하여 세포 증식을 측정하였다.
SDS-PAGE 및 크기-배제 크로마토그래피(SEC ; size-exclusion chromatography)에 의한 상기 정제된 항체들의 특정화는 상기 클론 1.109 경쇄를 제외하고는 전형적인 결과들을 산출하였으며, 이는 SDS-PAGE 상에서 기대된 것 보다 더 크게 이동하였다. N-연결 글리코실화 사이트 시퀀스가 앞서 CDR1에서 언급되었기 때문에 이러한 예외는 예기치 못한 것은 아니다. 기대된 것 보다 더 큰 이동은 이 글리코실화 사이트가 점거되었다는 것을 암시한다.
상기 항체들의 N-말단 시퀀싱은 신호 펩티드들이 기대된 되로 진행되었으며 상기 중쇄 N-말단 글루타민 잔기들이 기대될 수 있는 바와 같이 피로글루탐산(pyroglutamic acid)으로 유사하게 고리화되었다는 것을 확인하고 있다. 효소적 탈글리코실화(enzymatic deglycosylation) 후에 개개 항체 쇄들에 대하여 질량분석(Mass spectrometry)을 수행하였다. 상기 중쇄들의 질량들은 상기 N-말단 글루타민 잔기들이 피로글루탐산으로 고리화되었다는 것과 상기 C-말단 리신 잔기들이 부재라는 것을 확인하고 있다. 다른 전사후 변형들은 언급되지 않았다. 상기 클론 1.2 SM 및 2.360 경쇄들의 질량들은 이들이 전사후 변형들 없이 손상되지 않았다는 것을 확인하고 있다. 상기 클론 1.109 경쇄에 대한 질량은 수득되지 않았으며, 아마도 상기 글리코실화 사이트가 효소적 제거에 대해 저항성이었으며, 따라서 정확한 질량이 수득되지 않았을 것으로 여겨진다.
실시예 4 : 체세포 돌연변이(SMs)들의 보정
공지의 생식계열 시퀀스들에 대한 IgG2 클론 1.2, 1.109 및 2.360 항체들의 시퀀스 비교는 도 1a 및 도 1b들에 나타낸 바와 같은 성숙한 시퀀스에 대한 것인 표 11(가장 근접한 생식계열 시퀀스에 대한 체세포 돌연변이들)의 번호와 함께 표 11 내에 나타내듯이 다음의 체세포 돌연변이들을 밝혀냈다.
[표 11]
상기 체세포 돌연변이들이 생식계열 잔기들로 전환될 수 있는 지의 여부를 시험하기 위하여, 상기 적절한 구축물들을 생성시켰으며, COS/PKB 세포들 내에서의 일시적 발현에 의해 발현시켰으며, 단백질 A 크로마토그래피로 정제하였다. 이들 항체들을 IgG2 클론 1.2 SM, 1.109 SM 및 2.360 SM (SM = 체세포 돌연변이 큐어링된)으로 표기하였다. 상기 1.109 LC에 대하여는, 2개의 구축물들이 사용되었다. 첫번째의 구축물에 있어서, Asn-28을 Asp-28로 전환시켜 N-연결 글리코실화 사이트를 제거하고, 그리고 두번째의 구축물에 있어서, Asn-28을 Asp-28로 전환시키고, Asn-45를 Lys-45로 전환시켰다. 수율은 1.7 내지 4.5㎎/ℓ이었으며, 이는 이러한 발현 시스템에 대하여 기대되는 범위 이내이다.
SDS-PAGE 및 크기-배제 크로마토그래피(SEC)에 의한 상기 정제된 항체들의 특정화는 전형적인 결과들을 산출하였다. IgG2 클론 1.109 SM의 2개의 형태들의 SDS-PAGE는 경쇄가 부모 항체의 경쇄 보다 더 빨리 이동하였다는 것을 나타내어 상기 N-연결 글리코실화 사이트가 제거되었다는 것을 확인하고 있다. N-말단 시퀀싱은 상기 항체쇄들의 상기 N-말단이 손상되지 않았다는 것을 나타내었으며, 질량분석은 상기 체세포 돌연변이들이 생식계열 잔기들로 전환되었다는 것을 나타내었다.
실시예
5 : 체세포 돌연변이-보정 항체들의 특정화
상기 체세포 돌연변이들의 보정 후, 상기 정제된 IgG2 항체들을 상기 AML-5 및 사이노몰거스 골수 증식 분석에서 재시험하였다. 상기 AML-5 증식에서의 IC50은 부모 IgG2 항체들에 비하여 IgG2 클론 1.2 SM 또는 1.109 SM(SM = 체세포 돌연변이 큐어링된)에 대하여 변화되지 않았으나, 상기 IgG2 클론 2.360 SM 항체에 대한 포텐시에서는 10배의 손실이 있었다(표 12를 참조).
단량체성 c-fms 단백질로 체세포 돌연변이 보정된 항체들의 결합 친화도(binding affinities)들을 바이어코어 3000 기구(Biacore 3000 instrument)를 사용하여 표면플라스몬공명(surface plasmon resonance)에 의해 측정하였다. IgG2 클론 1.2 SM 항체의 친화도는 근본적으로 부모 항체들로부터 변화하지 않은 반면에, IgG2 클론 1.109 SM 및 2.360 SM 항체들의 친화도들은 개개 부모 항체들 보다 약 2배 정도 덜 하였다(표 12를 참조하시오).
125I-hCSF-1의 AML-5 세포들에의 결합을 저해하는 능력에 대하여 상기 부모(PT) 및 SM IgG2 항체들을 더 시험하였다. 먼저 125I-hCSF-1의 AML-5 세포들에의 뚜렷한 결합 친화도가 46pM이 되는 것으로 결정되었으며, 표지되지 않은 hCSF-1의 Kl은 17.8pM이었다(실시예 10을 참조). 표 12(부모(PT) 대 생식계열화된 항체들(SM)의 특성들)에 나타낸 바와 같이, 항체 1.2에 대한 Kl값은 상기 AML-5 생물검정에서의 IC50값과 일치하였으며, 1.2 SM은 유사한 결과들을 나타내었다. 항체 1.109에 대한 Kl값 역시 상기 IC50값과 일치하였으며, 바이어코어에 의해 측정된 바와 같은 단핵구성 c-fms에 대한 친화도에 있어서 2배의 손실이 있음에도 불구하고 상기 1.109 SM 항체에 대하여 변화가 없었다. 항체 2.360은 항체 1.2 및 1.109와 마찬가지로 저해하지 않았으며, 또한 2.360 SM은 그 부모 항체 보다 덜 저해하였다.
[표 12]
인간 또는 사이노몰거스 골수 단핵구성 세포들을 사용하는 증식 분석들에서 상기 1.2 SM 항체의 활성을 더 조사하였다. 인간 분석에 대하여는, 감소하는 농도들의 항체 1.2 SM의 존재 중에서 인간 세포들을 11.1ng/㎖의 재조합 인간 CSF-1과 함께 배양시켰다. 상기 사이노몰거스 분석에 대하여는, 감소하는 농도들의 항체 1.2 SM의 존재 중에서 사이노몰거스 세포들을 29.63ng/㎖의 재조합 인간 CSF-1과 함께 배양시켰다. 인간 IgG2 항체를 대조 실험들에서 사용하였다. 7일 후, 셀타이터-글로(CellTiter-Glo ; 미합중국 위스콘신주 매디슨 소재 프로메가(Promega))를 사용하여 세포 증식을 측정하여 ATP의 수준들을 결정하였다. 비선형의 회귀곡선 피트(nonlinear regression curve fit)를 수행하여 상기 항체의 IC50을 결정하였다. 표 13(세포 증식 분석에서의 클론 1.2 SM의 활성)은 실험들의 3개의 세트들의 결과들을 나타내고 있다.
[표 13]
실시예 6 : c-fms 티로신 인산화의 저해
항-c-fms IgG2 mAbs, 1.109, 1.2 및 2.360들이 포스포티로신(pTyr)-반응을 완전하거나 또는 거의 완전한 저해할 수 있다는 것을 입증하기 위하여, CSF-1 자극에 앞서 293T/c-fms 세포들을 이들 mAbs들로 1시간 동안 여러 농도들에서 37℃에서 처리하였다.
적정희석(titration dilutions)들을 사용하는 상기 IgG2 mAbs의 여러 농도들은 1.0, 0.1, 0.01, 0.001 및 0.0001㎍/㎖이었다. 대조로서, 차단되지 않은 항-c-fms 모노클로날 항체, mAb 3-4A4(바이오소스 인터내셔널(BioSource, Intl.)) 및 비-상관의 항체 hCD39 M105들을 각각 1.0㎍/㎖로 사용하였다. 혈청-결핍 293T/c-fms세포들을 앞서 언급한 여러 농도들을 사용하는 IgG2 (PT) mAbs 각각으로 처리하였으며, 각 농도는 50ng/㎖에서 5분간의 CSF-1 자극 5분 전에 10배의 희석으로 변화시켰다. 자극 후, 전체 세포 용해물들을 수집하고, 항-c-fms C20 폴리클로날 항체(산타크루즈 바이오테크놀로지스, 인코포레이티드((Santa Cruz Biotechnology, Inc.))와 함께 4℃에서 밤새도록 면역침강시켰으며, 웨스턴블럿으로 검사하였으며, 여기에서 블럿(blot)을 각각 c-fms의 티로신 인산화 및 c-fms 자체의 수준들에 대하여 일반 항-pTyr 항체, 4G10(업스테이트 바이오테크놀로지(Upstate Biotechnology) 및 항-c-fms C20 항체)들로 면역탐침(immunoprobed) 시켰다.
24X10㎝ 접시들 상에서 37℃, 5% 이산화탄소(CO2) 내에서 상기 293T/c-fms 세포들을 성장시키기 위하여, 11개의 T175 플라스크(약 50 내지 60% 융합성(confluent))들을 4㎖ 트립신/플라스크(기브코-인비트로겐(Gibco-Invitrogen))을 통하여 수집하였으며 70㎖ DMEM(기브코)/10% FBS(제이알에이취 바이오사이언시즈(JRH Biosciences)에로 전달시켰다. 계속해서 각 10㎝ 접시에 10㎖ 배지를 공급하고 그리고 상기 수집된 세포들 2㎖를 접종시켰다. DMEM/-FBS 배지를 37℃에서 1시간 동안 준비하였다. 1신중한 흡기를 통하여 10㎝ 접시들로부터 배양 배지를 제거하였으며, 마찬가지로 FBS-함유 배지를 가능한 한 제거하였다. 10㎖ DMEM/-FBS를 첨가하고 그리고 그 혼합물을 37℃에서 1시간 동안 배양시켰다.
37℃에서 1시간 동안 혈청결핍시킨 후, 상기 배지를 제거하였다. 항체 처리 및 마이너스-Ab(minus-Ab) 대조들을 연속적으로 희석시킨 Ab-함유 샘플들 또는 DMEM/-FBS 단독의 둘 중 하나의 4.0㎖와 함께 설정하였으며, 다시 37℃에서 1시간 동안 더 배양시켜 총 2시간의 혈청결핍이 제공되도록 하였다. 상기 항체 전-처리 및 리간드 자극을 표 14에 나타내었다.
[표 14]
50ng/㎕에서 CSF-1(알앤디 시스템즈(R&D Systems)/216-MC/롯트번호 CC093091)의 새로운 바이알(25㎍/바이알)을 500㎕ PBS(기브코)/0.1% BSA(소혈청알부민, 시그마) 내에서 재구성하였으며, 얼음상에서 유지시켰다. Ab-처리의 끝에서 대략 5분 전에 CSF-1 스톡(stock)(60㎕)의 1:1000 희석을 준비하여 DMEM/-PBS로 투입하였다. 293T/c-fms 세포들을 50ng/㎖의 CSF-1과 함께 37℃에서 5분간 배양시켰다. 그 상청액들을 제거하고 그리고 2㎖의 차가운 용해물 완충제(lysis buffer)(100㎖의 PBS/1% 트리톤, 100㎕의 0.5M EDTA, 100㎕의 1.0M NaF, 200㎕의 0.5M 베타-글리세롤포스페이트, 500㎕의 바나듐산나트륨(sodium vanadate)(100배), 10.0㎕의 오카다산(okadaic acid)(10,000배) 및 4정의 완전한 프로테아제 저해제))를 첨가하였다. 용해물(2.0㎖)을 30㎕의 50% 단백질 A/G 세파로스(Protein A/G Sepharose ; 아메르샴(Amersham))과 결합시키고, 록커 플랫폼(rocker platform) 상에서 4℃에서 1시간 동안 배양시켜 비-특이적인 결합 단백질들을 프리클리어링(pre-clear)시켰다. 분획(fractions)들을 회전시킨 후, 그 상청액들을 새로운 15㎖의 시험관에 딸아내었다.
전체 세포 용해물들을 항-c-fms C20(25㎕; 0.2㎍/㎕)로 면역침강 시켰다. 상기 항체-세포 용해물 혼합물들을 록커 플랫폼 상에서 4℃에서 밤새도록 배양시켜 총 c-fms에 대하여 조사하였다. 당나귀 항-토끼 IgG/HRP( 차단용액 내 1 : 10,000 ; 잭슨(Jackson))를 첨가하고 계속해서 4℃에서 30분 더 배양시켰다. 상기 면역착물(immunocomplexes)들을 SDS-PAGE 상에서 지속시키고 그리고 면역블롯팅(immunoblotted) 시켰다. 웨스턴블롯들을 pTyr c-fms 및 총 c-fms 각각들의 검출을 위하여 항-pTyr 4G10 또는 항-c-fms C20 둘 중의 어느 하나에 대하여 조사하였다.
도 5에 나타난 바와 같이, IgG2 클론들(PT) 1.109, 1.2 및 2.360들은 293T3/c-fms 분석 시스템에서 리간드-유도 pTyr/c-fms를 저해하는 능력을 나타내었다. CSF-1 자극 한 시간 전에의 IgG2 클론(PT) 1.109 또는 1.2들의 0.1㎍/㎖(8.3nM)로의 처리는 포스포티로신 시그널(phosphotyrosine signal)을 배경 수준(background levels)들로 감소시켰다. 반면에, IgG2 클론(PT) 2.360은 1.0㎍/㎖(8.3nM)에서 동등한 저해를 생성하였다. 그러나, 0.01㎍/㎖(0.83nM) 또는 그 이하에서의 항체들의 처리는 pTyr 저해라는 결과를 나타내지 않았다. 대조적으로, 비-차단된 항-c-fms 3-4A4 및 비-연관의 hCD39 M 105 항체들은 가장 높은 투여량(1.0㎍/㎖)에서 -mAb/+CSF-l 대조에 비하여 리간드-유도 pTyr 시그널에 대하여 영향을 주지 않았다. 따라서, pTyr 형성의 저해는 CSF-1 결합에 직접적으로 연관된다.
이들 분석들 내에서 사용된 상기 293T/c-fms 트랜스펙턴트(transfectants)들이 앞서 측정된 약 30,000수용체들/세포의 세포-표면 c-fms 밀도를 유지한다고 가정하면, 약 3만개의 세포들이 0.1㎍/㎖에서의 4.0㎖ 전처리에서의 약 5.0*1011 mAb 보다 현저하게 적은 약 90*106 c-fms들을 떠 맡을 수 있다. 표적에 비해 약 10,000배 과량으로 존재하는 mAb는 마찬가지로 획득가능한 c-fms의 포화(saturation)를 만든다. 이는 8.3nM 클론(PT) 1.109 또는 1.2가 50ng/㎖ 또는 1.0nM에서 또는 대략 10:1(mAb:c-fms) 몰비(molar ration)에서의 CSF-1의 시그널링을 효과적으로 차단한다는 것을 나타내고 있다. 클론(PT) 1.109 및 1.2에 대한 유효성의 문턱값은 유사하게 이 분석 시스템에서 0.1 내지 0.01㎍/㎖(0.83 내지 8.3nM) 사이에 떨어지게 된다.
CSF-1 부가의 부재 중에서의 1㎍/㎖의 IgG2(PT) mAbs로의 처리는 배경수준들 이상의 pTyr 시그널을 생성하지 않았다. 10㎍/㎖에서 사용된 3개의 IgG2(PT) 형태들 모두에 대한 앞서의 실험들은 또한 동일한 조건들 하에서 pTyr 시그널들을 나타내지 못하였다. 이들 mAbs들과 연관된 항진 활성은 측정되지 않았다.
실시예
7 :
IgG
2
PT
및
SM
형태들을 사용하는
리간드
-유도
pTyr
/c-
fms
의 저해
이 연구의 목적은 그들의 개별적 부모형들(PT)과 비교하여 IgG2 클론들 1.109, 1.2 및 2.360의 생식계열-역전된(SM) 형태들에서 어떠한 기능적 변화들이 있는 지의 여부를 결정하는 것이다. 본 실험을 위한 293T/c-fms 세포들을 준비하기 위하여, 5개의 T175(약 80 내지 90% 융합성)로부터 성장하는 세포들을 4㎖ 트립신/플라스크를 통하여 수집하였으며, 10% FBS를 갖는 75㎖의 DMEM에로 옮겼다. 개개 24X10㎝ 접시들에, 9㎖의 배지를 첨가하고 그리고 3㎖의 수집된 세포들을 접종시켰다. DMEM/-FBS 배지를 준비하거나 및/또는 37℃에서 1시간 동안 가온시켰다. 신중한 흡기를 통하여 10㎝ 접시들로부터 배양 배지를 제거하여 FBS-함유 배지를 가능한 한 제거하였다. DMEM/-FBS(10㎖ )를 첨가하고 그리고 그 혼합물을 37℃에서 1시간 동안 배양시켰다. 차가운 용해물 완충제를 준비하고 얼음 상에서 유지시켰다.
표 15(IgG2 c-fms 모노클로날 항체들의 적정)에 기술한 바에 따라 모노클로날 항체 적정들을 준비하고 그리고 실온에서 유지시켰다.
[표 15]
일련의 연속하는 항체 희석물들(300㎕ + 2.7㎖ DMEM/-FBS)을 각 mAb에 대하여 1.0㎍/㎖ 내지 0.1㎍/㎖의 범위 내에서 시험하였다. 37℃에서의 혈청결핍 1시간 후, 상기 배지를 제거하고 그리고 항체 전처리 및 마이너스-Ab들을 표 13에서 기술한 바와 유사하게 제조하였다. 상기 항체 전처리 및 리간드 자극을 이하의 표 16에 기술하였다.
[표 16]
항-c-fms mAbs(SM 형태들)에 의한 리간드-유도 pTyr을 상기 PT형태들에 대하여 실시예 6에서 기술한 바와 같이 수행하였다.
1.0 및 0.1㎍/㎖에서의 상기 3개의 IgG2 mAbs들의 형태들에 대한 PT의 효과들을 비교하기 위하여 상기 293T/c-fms 세포들을 사용하는 실험들은 리간드-유도 pTyr/C-fms를 저해하는 능력에 있어서 차이점들이 없음을 나타내고 있다(도 6을 참조). 클론 1.109 및 1.2(둘 다 PT 및 SM 형태들)들은 2.360(PT 및 SM 형태들)에 대한 것 보다 더 낮은 농도들에서 저해를 나타내었다.
50ng/㎖(1.0nM)에서의 CSF-1의 첨가에 앞서 293T/c-fms 세포들을 0.1㎍/㎖(8.3mM)로 처리하는 경우, 클론 1.109 및 1.2(PT 또는 SM)들은 시험관 내에서 리간드-유도 pTyr/c-fms를 방지할 수 있다. pTyr/c-fms의 형성을 차단하는 이들 모노클로날 항체들의 능력은 CSF-1 시그널링, 단핵구 이동 및 후속적으로 TAMs들의 축적으로 유도될 수 있다. 어떠한 항진 활성도 이들 mAbs들이 비-CSF-1 의존적인 방법에서 상기 수용체를 활성화시키는 것을 피하도록 하는 것과 연관되는 것으로 나타나지는 않았다. 1.0㎍/㎖ 및 10㎍/㎖과 같은 높은 농도에서 사용되는 경우, 상기 mAbs들은 항진 활성을 나타내지 않았다(데이터는 나타내지 않음).
따라서, 상기 mAbs들은 시험관 내에서 리간드-유도 pTyr/c-fms를 방지할 수 있다.
실시예
8 : 항-c-
fms
mAbs
에 의한 c-
fms
의
면역침강
c-fms를 결합하고 그리고 면역침강시키는 IgG2 anti-c-fms mAbs의 능력은 앞서 기술한 바와 같이 안정적으로-형질전환된 293T/c-fms 세포들을 사용하는 것에 의하여 달성되었다. 자극되지 않은 전체 세포 용해물들을 mAb(PT 및 SM) 및 항-c-fms C20 항체들 각각과 함께 밤새도록 면역침강시켰으며, c-fms의 검출을 위한 탐침으로서 C20 Ab(산타크루즈 바이오테크놀로지스, 인코포레이티드)와 함께 웨스턴블럿으로 검사하였다. c-fms는 모노클로날 항체들에 의하여 면역침강되었다. 37℃/5% CO2에서 약 75% 융합성까지 성장시킨, 안정적으로 형질전환된 293T/c-fms의 용해물들을 준비하고 그리고 표 17에 나타낸 바와 같이 모노클로날 항체들과 결합시켰다.
[표 17]
안정한 293T/c-fms의 미처리된 전체 세포 용해물들을 사용하는 면역침강 실험들은 폴리클로날 항-c-fms C20 대조와 비교하여 여러 mAbs(2.360 SM은 제외)들이 c-fms와 결합하고 그리고 침강시키는 상당한 능력을 입증하였으나, 그러나, 클론 2.360-SM은 이 분석에 있어서 감소된 용량(capacity)을 나타내었다(도 7을 참조).
실시예
9 :
IgG2
mAbs
들에 의한
SNP
-변종들의
면역침강
단일 뉴클레오티드 다형성(polymorphisms)들 또는 SNPs들은 게놈 시퀀스 내의 단일 뉴클레오티드(A, G, T 또는 C)가 변화되는 경우에 발생하는 DNA 시퀀스 변이들이다. SNPs들은 인간 게놈의 코딩 영역들 및 비-코딩 영역들 둘 다에서 발생할 수 있다. 많은 SNPs들은 세포 기능에 대해 영향을 주지 않으나, 과학자들은 다른 SNPs들이 사람들이 질병에 걸리게 하거나 도는 사람들의 약물 반응에 영향을 줄 수 있을 것이라고 여기고 있다. DNA 시퀀스 내에서의 변이들은 사람이 질병, 환경적 자극들(예를 들면, 박테리아, 바이러스, 독소 및 화학약품들), 약물 및 다른 요법들에 대해 어떻게 반응하는 지에 대한 주요한 영향을 미칠 수 있다. 이러한 이유로, SNPs들은 생물의공학적 연구(biomedical research), 약제학적 제품 개발 및 의학적 진단에서 큰 가치가 있다. 더욱이, 과학자들이 암, 당뇨병 및 혈관 질병(vascular diseases) 등과 같은 복합질병(complex diseases)와 연관되는 동의유전자(multiple genes)들의 동정에서 SNP 맵(maps)들의 작성을 가능하게 할 수 있다.
인간 c-fms의 세포외 영역은 5개의 면역글로블린(Ig)-형의 반복되는 도메인들(A 내지 E들로 표기됨)로 분할될 수 있다. 예를 들면, 함페 에이(Hampe, A.)와 그의 동료들의 (1989) Oncogene Res. 4:9-17 for a discussion of the human domains를 참조하시오. 생쥐 단백질 내에서의 대응하는 도메인들에 대하여는, 예를 들면, 왕(Wang)과 그의 동료들의 (1993) Molecular and Cell Biology 13:5348-5359을 참조하시오. 도메인 A 내지 C들은 상기 CSF-1 결합 영역을 포함하는 것으로 나타난 반면에, 도메인 D는 리간드의 결합에 대한 수용체 이량체화(receptor dimerization)을 조절하는 것을 돕는 것으로 나타났다. 인간 c-fms의 3개의 자연적으로 발생하는 SNP-변이들 즉 A245S, Ig-도메인 C 내의 V279M 및 Ig-도메인 D 내의 H362R들이 제조되었다(상기 c-fms의 세포외 도메인의 아미노산 시퀀스에 대해서는 도 8을 참조하시오). 이들 SNPs들은 상기 CSF-1 결합 영역 내 또는 근처에서 발견되었으며, 웨스턴블럿들에 의한 조사로 항-c-fms H-300(인간 c-fms/CSF-1R의 N-말단 근처에서 맵핑되는 아미노산들 11-310에 대해 야기된 토끼 폴리클로날 항체 ; 산타크루즈 바이오테크놀로지스, 인코포레이티드 Cat. No. sc-13949)에 대해 입증되었다.
인간 c-fms SNP 변이들이 어떻게 본 명세서에서 제공되는 여러 c-fms Abs들과 상호작용하는 지를 연구하기 위하여, 앞서 논의한 바와 같은 c-fms SNP 변이들의 3가지 타입들을 발현하는 일시적으로 감염된 293T 세포들 및 야생형(WT) c-fms(마찬가지로 무관한, 벡터-매칭된 대조)들을 사용하여 면역침강을 통한 SNP-변이들에 결합하는 항-c-fms mAb 각각의 능력을 평가하였다.
293T 세포들을 c-fms A245S, V279M, H362R, WT c-fms 및 포유동물 발현 벡터 pCIneo 내의 무관한 대조 구축물들을 갖는 이중의 10-㎝ 접시들에 감염시켰으며, 또한 37℃/5% CO2에서 48시간 동안 성장시켰다. 세포 용해물들을 앞서 기술한 바와 같이 준비하였다. 1.0㎖ 분획(aliquots)들 내의 2.5㎍/㎖의 항-c-fms mAbs 및 폴리클로날 항-c-fms C20 또는 항-c-kit C19들을 표 18에 나타낸 바와 같이 각 용해물에 첨가하였다.
[표 18-1]
[표 18-2]
상기 세포들을 실시예 6에서 기술한 바와 같이 록커 상에서 4℃에서 밤새도록 배양시켰다.
상기 항체들은 야생형(WT) 대조에 비해 SNP 형태들에 결합하는 능력의 손실이 없었음을 나타내었다(도 9). 상기 mAbs들은 자연적으로 발생하는 범위의 c-fms 변이들에 결합하는 능력을 갖는 것으로 나타나고 있다.
폴리클로날 항-c-fms 대조에 비해 안정한 293T/c-fms의 미처리된 전체 세포 용해물들이 c-fms에 결합하고 그리고 침강하는 여러 mAbs들(2.360 SM은 제외)의 능력과 동등함을 입증하고 있으며; 클론 2.360 SM은 이 분석에서 명백하게 감소된 능력을 나타내었다. 일시적으로-감염된 293T/c-fms 세포들로부터의 c-fms 및 SNP 변이들을 면역침강시키는 여러 mAbs들의 능력에 대한 검사에서 상기 SNP 형태들에 경합하는 능력의 손실이 없는 것으로 나타났다. c-fms SNPs들에 결합하는 여러 mAbs들의 능력은 이들이 인간들에 대해 존재하는 것으로 알려진 변이들의 스펙트럼을 가로질러 c-fms 단백질들을 인식한다는 것을 나타내었다.
실시예 10 : 항-c-fms mAbs에 의한
125
I-hCSF-1 결합의 저해
세포 표면 발현 인간 c-fms에 대한 항-c-fms mAbs의 친화도는 AML-5 세포들에의 125I-hCSF-1 결합의 저해를 측정하는 것에 의해 결정되었다.
재조합 hCSF-1(암겐)을 125I(아메르샴) 및 아이오도-젠®(IODO-GEN® ; 피어스(Pierce))들을 사용하여 요오드화시켰다. 75㎕의 PBS, 10㎍의 hCSF-1 및 1mCi(밀리퀴리)의 125I들을 아이오도-젠® 사전코팅된 요오드화 시험관(iodination tube)에 첨가하고 그리고 얼음 상에서 15분간 방치시켰다. 상기 혼합물을 평형화된(equilibrated) 2㎖의 P6 컬럼으로 옮겼으며, 여기에서 125I-hCSF-l을 겔여과에 의해 유리 125I로부터 분리시켰다. 요오드화된 hCSF-1을 포함하는 분획들을 모았으며, 계속해서 결합 배지(binding media ; 2.5% 소알부민 분획 V, 20mM의 헤페스 및 0.2%의 소듐아지드(sodium azide)를 갖는 RPMI-1640, pH 7.2) 내에서 100nM의 농도로 희석시켰다. hCSF-1의 초기 단백질 농도 및 125I-hCSF-1의 분획이 별도의 125I가 생략된 요오드화 프로토콜을 통과한 대조 실험으로부터의 80%의 회수(recovery)에 기초하여 4.8*1015cpm/mmol의 특이적인 활성이 산출되었다.
AML-5 세포들의 표면 상에 발현된 c-fms에 결합하는 cCSF-1에 대한 KD 및 K1을 결정하기 위하여 각 저해 분석(inhibition assay)과 함께 포화 방사성리간드 결합 실험을 수행하였다. 혼합물들을 150㎕/웰의 총 용적을 갖는 96-웰(96-well) 둥근바닥 마이크로타이터 접시들 내에 혼합물들을 투입하였다. 모든 시약들을 0.2% 소듐아지드를 포함하는 결합 배지 내에서 희석시켰으며, 실험들은 4℃에서 수행하여 잠재적인 수용체의 내재화(internalization) 및 흘리기(shedding)를 최소화시켰다.
포화 결합 분석에 대하여, 125I-hCSF-1을 1.7nM에서 시작하여 일련의 2배 희석시켰으며, 12개의 웰들에로 약 1.5pM의 농도까지 진행시켰다. 1.000배의 몰의 과량의 표지화되지 않은 hCSF-1의 존재 중에서 125I-hCSF-1(약 80pM, 3배(triplicate))의 단일 농도에서 비-특이적인 결합이 측정되었으며, 방사성표지된 hCSF-1의 농도의 선형함수(linear function)가 존재하는 것으로 추측되었다.
125I-hCSF-1 저해 분석에 대하여는, 표지되지 않은 hCSF-1이 5nM의 개시 농도에서 착수되었다. 항-c-fms 1.2, 1.109 및 2.360(PT 및 SM 각각에 대하여)들에 대한 개시 농도들은 각각 0.312nM, 1.25nM 및 20nM 이었다. 각 샘플을 15웰들에 연속적으로 2배 희석시켰다. 결합 배지 단독의 3중의 웰들 및 저해 백분율을 결정하기 위한 대조로서 1,000배의 몰의 과량의 표지화되지 않은 hCSF-1의 3중의 웰들을 상기 분석의 시작, 중간 및 끝에서 구축하였다. 125I-hCSF-1의 단일의 농도(약 9pM)를 각 웰이 첨가하였다.
AML-5 세포들을 PBS로 2회 세척하고 그리고 배양 직전에 1*105세포/웰로 각 분석 플레이트에 첨가하였다.
두 분석들을 소궤도 진탕기(miniorbital shaker) 상에서 4℃에서 시간 경로 실험(time course experiments)들에서 결정된 바와 같이 평형에 도달하는 데 필요한 시간의 길이인 4시간 동안 배양시켰다. 각 배양 혼합물의 2개의 60㎕ 분획들을 200㎕의 프탈레이트 오일(phthalate oil)을 포함하는 냉각된 400㎕ 폴리에틸렌 원심분리관들로 옮기고 4℃의 탁상 마이크로퓨즈(tabletop microfuge)(소르볼(Sorvall) 내에서 9615*g에서 1.5분간 회전시켜 유리 125I-hCSF-1으로부터 세포 연관 125I-hCSF-1을 분리시켰다. 오일관들을 절단하고, 각 세포 펠릿 및 상청액을 개개 12*75㎜ 유리관들 내에 수집하고 그리고 cpm 측정을 위하여 코브라 감마 계수기(COBRA gamma counter ; 팩커드 인스트루먼트 컴퍼니(Packard Instrument Company)에 탑재하였다. 각 웰로부터 취하여진 이중의 분획들로부터의 cpm을 평균하여 분석하였다.
포화 결합 데이터(saturation binding data)들은 프리즘 버전 3.03(Prism version 3.03(그라패드 소프트웨어 인코포레이티드(GraphPad Software, Inc.))에서의 비선형 회귀를 통한 단순한 1-사이트결합 등식에 들어맞아서 뚜렷한 세포 표면 발현 인간 c-fms에의 125I-hCSF-1 결합에 대하여 46pM의 평균 KD를 수득하였다. 저해 데이터(inhibition data)들은 동시발생의 결합 분석에서 얻어지는 125I-hCSF-1에 대한 KO값을 사용하는 프리즘에서의 비선형 회귀를 통한 단순한 사이트경쟁저해식(site competitive inhibition equation)에 들어맞아서 각 항-c-fms mAb에 대하여와 마찬가지로 표지화되지 않은 hCSF-1(명백한 평균 K1 = 17.8pM)에 대한 K1값을 생성하였다. 2개의 실험들로부터의 평균 K1값들이 보고되었다(표 13을 참조).
실시예 11 : 항-c-fms mAbs들에 결합하는 단량체성 c-fms에 대한 속도 및 친화성 상수들의 결정
항-c-fms mAbs에 대한 인간 c-fms (l-512).pHIS(암겐)의 친화성을 바이어코어에 의해 측정하였다. 실험들은 CM4 센서칩(CM4 sensor chip)을 장착한 바이어코어 3000 기구(바이어코어 에이비)를 사용하여 25℃에서 수행되었다. 센서칩들, 아민 커플링 시약(amine coupling reagents ; EDC(1-에틸-3(3-디메틸아미노프로필)-카르보디이미드 하이드로클로라이드), NHS(N-하이드록시숙신이미드) 및 에탄올아민-염산, pH 8.5), 10mM 아세트산나트륨, pH 5.5, HBS-EP(0.01M HEPES pH 7.4, 0.15M 염화나트륨, 3mM EDTA, 0.005용적/용적% 계면활성제 P20) 및 10mM 글리신-염산, pH 1.5들을 바이어코어 에이비로부터 구입하였다. 소혈청알부민(BSA, 보부미나르 표준 분말(Bovuminar Standard Powder))을 세롤로지컬 코포레이션(Serological Corporation)으로부터 구입하였다. Fcγ 단편 특이적인 어피니퓨어 고트 항-인간 IgG(AffiniPure Goat Anti-Human IgG)를 잭슨 임뮤노리서치 라보레토리즈(Jackson ImmunoResearch Laboratories)로부터 구입하였다.
항-인간 IgG, Fcγ 특이적 포획 항체(anti-human IgG, Fcγ specific capture antibody)를 런닝버퍼(running buffer)로서 HBS-EP를 갖는 표준 아민-커플링 화학(standard amine-coupling chemistry)을 사용하여 CM4 칩에 공유적으로 부동태화시켰다. 간단하게, 각 플로우셀을 5㎕/분의 흐름속도에서 0.1M NHS 및 0.4M EDC의 1 : 1(용적/용적) 혼합물로 7분간 활성화시켰다. 10mM 아세트산나트륨 내의 28㎍/㎖의 농도의 염소 항-인간 IgG, pH 5.5를 약 2700RUs의 밀도에서 부동태화시켰다. 잔존의 반응성 표면들을 5mM/분에서 1M 에탄올아민의 7분간의 주사로 비활성화시켰다. 100㎕/분에서의 10mM 글리신 염산, pH 1.5의 3개의 50㎕ 주사들을 사용하여 임의의 잔존하는 비공유적으로 결합된 포획 항체를 제거하고 그리고 각 표면을 조정하였다. 모든 잔류하는 단계들에 대하여 상기 런닝버퍼를 0.1㎎/㎖ BSA를 갖는 HBS-EP로 교환하였다.
0.25㎍/㎖의 항-c-fms 1.2 또는 1.2 SM을 10㎕/분에서 2분간 하나의 플로우셀 내의 염소 항-인간 IgG, Fcγ 상으로 주사하여 약 47RUs의 표면밀도를 수득하였다. 염소 항-인간 IgG, Fcγ 단독을 갖는 다른 플로우셀을 대조 표면으로 사용하였다. 각 분석은 시그널을 안정화시키기 위한 검출대상물로서 10회의 완충제로 개시시켰다. 인간 단량체성 c-fms (l-512).pHIS 샘플들을 30, 10, 3.33, 1.11, 0.37 및 0.12nM의 농도로 3중으로 준비하였으며, 6개의 버퍼 블랭크(buffer blanks)들과 함께 무작위적인 순서로 100㎕/분으로 포획된 항-c-fms 및 대조 표면들 둘 다 위로 주사하였다. 각 복합체를 2분간 결합시키고(associate) 그리고 5분간 분리시켰다(dissociate). 각 c-fms 또는 버퍼 주사를 10mM 글리신 염산, pH 1.5를 100㎕/분의 30-초의 펄스(pulse)로 주사하고 30초의 버퍼의 주사를 한 후 상기 표면들을 재생시켰다.
다른 항-c-fms 항체들을 유사한 방법으로 시험하였으나, 결합 특성들에서의 차이를 고려하여 프로토콜(protocol)을 변화시켰다. 항c-fms 1.109 및 1.109 SM들을 각각 염소 항-인간 IgG 상에 0.5㎍/㎖로 10㎕/분으로 1.5분간 주사하여 각각 59 및 91RUs의 표면밀도들을 수득하였다. 항-c-fms 1.109 결합에 대하여 인간 c-fms (1-512).pHIS 샘플들을 10, 3.33, 1.11, 0.37, 0.12 및 0.041nM의 농도들로 준비하였으며, 항-c-fms 1.109 SM 결합에 대하여는 0.041nM 샘플을 제외하고는 동일하게 구축하였다. 인간 단량체성 c-fms (l-512).pHIS를 20분간 항-c-fms 1.109로부터 그리고 1.109 SM을 15분간 분리되도록 하였다. 항 c-fms 2.360 및 2.360 SM들을 각각 1㎍/㎖의 염소 항-인간 IgG, Fcγ 상으로 각각 10㎕/분으로 1.5 또는 2분간 주사하여 약 100RUs의 표면밀도들을 수득하였다. 항-c-fms 2.360 결합에 대하여 c- fms (l-512).pHIS 샘플들을 30, 10, 3.33, 1.11 및 0.37nM의 농도들로 주사하였으며, 항-c-fms 2.360 SM 결합에 대하여는 0.12nM을 추가하여 동일하게 구축하였다. 인간 단량체성 c-fms (l-512).pHIS를 8분간 항-c-fms 2.360로부터 그리고 5분간 2.360 SM으로부터 분리되도록 하였다.
규모의 굴절율 변화(bulk refractive index)의 변화를 제거하기 위하여 대조 표면 반응들을 차감하고, 그리고 계속해서 실험적인 플로우셀들로부터 체계적인 인공물들(systematic artifacts)을 제거하기 위하여 평균된 버퍼 블랭크 반응을 차감하는 것에 의하여 데이터를 이중으로 대조하였다. 상기 데이터를 처리하고 그리고 바이어이벨류에이션(BIAevaluation) 내의 로컬 Rmax를 갖는 1 : 1 인터랙션 모델(버전 4.1, 바이어코어 에이비)에 맞추어 동력학적 속도 상수(kinetic rate constants)들 Ka 및 Kd들을 수득하였다. 비록 c-fms의 각 농도에서의 3중의 샘플들로부터 데이터들이 수집되기는 하였으나, 바이어이벨류에이션 소프트웨어에 고유한 변수의 개수 제한들로 인하여 단지 2중의 샘플들로부터의 데이터를 분석하였다. 상기 KD는 Kd/Ka의 몫으로부터 산출하였다. 그 결과들을 표 19(바이어코어 3000에 의해 측정된 바와 같은 가용성 단량체성 cc-fms에 대한 항-c-fms mAbs의 결합 친화성)에 나타내었다. 본 명세서에서 제공된 여러 샘플들로부터의 데이터를 표 20(mAbs 1.2, 1.109 및 2.360의 요약)에 요약하였다.
[표 19]
| 모노클로날 항체 |
AML-5 증식 IC50(pM)* |
사이노몰거스 골수 증식IC50(pM) |
AML-5세포들에의 125I-CSF-1의 결합의 저해(KI) | 바이어코어에 의한 c-fms에의 결합 친화성(KD)(pM) | 야생형 c-fms 및 SNPs를 발현하는 293T 세포들로부터의 IP | c-fms의 pTyr의 리간드 유도에 대한 저해 |
| 1.2 | 27 | 78 | 8.5 | 516 | +++ | +++ |
| 1.2 SM** | 12 | 81 | 11.5 | 548 | +++ | +++ |
| 1.109 | 27 | 16 | 13.5 | 51 | +++ | +++ |
| 1.109 SM** | 23 | 9.7 | 102 | +++ | +++ | |
| 2.360 | 60 | 67 | 약160 | 535 | +++ | ++ |
| 2.360 SM** |
약900 | 1200 | ++ | ++ | ||
| * 인간 표적에 대한 1차 세포 분석; **SM = 체세포 돌연변이 제거됨 | ||||||
실시예 12 : 항 c-fms 항체들 IgG
2
클론 1.109, 1.2 및 2.36들의 에피토프 매핑
c-fms 아비딘 융합 구축물들의 제조
상기 c-fms 아비딘 융합 발현 구축물들을 도 10에 나타내었다. 각 융합 단백질을 발현시키기 위하여, 인간 c-fms 세포외 도메인에 대한 코딩 시퀀스를 PCR증폭시켰으며, pCEP4-아비딘(N) 내로 복제시켜, 제한효소 XhoI를 사용하여 상기 c-fms 시퀀스가 치킨 아비딘 시퀀스(chicken avidin sequence)의 C-말단에 연결되도록 하였다. 치킨 아비딘에 대한 시그널 시퀀스가 pCEP4-아비딘(N) 벡터 내에 손상되지 않은 채로 잔류하였기 때문에 c-fms 시그널 시퀀스는 포함되지 않았다.
앞서 언급한 바와 같이, 상기 세포외 도메인은 5개의 서로 다른 Ig-형영역들을 포함한다. 인간 c-fms 내의 상기 서로 다른 도메인들은, 예를 들면, 함페(Hampe)와 그의 동료들의 1989, Oncogene Res. 4:9-17에 기술되어 있다. 생쥐 c-fms 내의 대응하는 도메인들의 논의에 대하여는, 예를 들면, 왕과 그의 동료들의 1993, Molecular and Cell Biology 13:5348-5359를 참조하시오. 아래의 서로 다른 아비딘 구축물들을 표시된 Ig-형 도메인들과 대응하도록 하여 제조하였다(상기 세포외 도메인의 아미노산 시퀀스 SEQ ID No 1에 대하여는 도 8을 참조하시오).
시그널 : 아미노산 1-19
Ig-형 1 도메인 : 아미노산 20-126
Ig-형 1-2 도메인 : 아미노산 20-223
Ig-형 1-3 도메인 : 아미노산 20-320
Ig-형 1-4 도메인 : 아미노산 20-418
Ig-형 1-5 도메인 : 아미노산 20-512
Ig-형 2 도메인 단독 : 아미노산 85-223.
따라서, 에피토프 매핑을 위한 인간 c-fms(끝이 절단된)의 특이적인 영역들을 생성하기 위해서는, 하기의 아미노산들을 표적으로 하는 PCR 증폭들을 수행하였다 : Ig-형 루프2 단독(TEPG-AQIV)과 마찬가지로, Ig-형 루프l(IPVI-ALLP), Ig-형 루프1 및 2(IPVI-AQIV), Ig-형 루프 1 내지 3(IPVI-EGLN), Ig-형 루프 1 내지 4(IPVI-GTLL) 및 Ig-형 루프 1 내지 5(IPVI-PPDE). 상기 어구에서 확인된 시퀀스들은 상기 도메인들 각각에 대하여 각각 개시 및 종결 시퀀스를 나타내고 있다(도 8을 참조하시오). 상기 도메인들의 본래의 3차원적인 구조를 유지하는 데 있어서 이황화 결합들이 중요하기 때문에, 표시된 특정의 영역들을 선택하여 이황화결합 형성에 포함되는 시스테인 잔기들을 보호하였다. 더욱이, Ig-형 루프 2 단독에 대한 상기 구축물은 동일한 이유로 상기 Ig-형 루프 1 유래의 일부 시퀀스들을 포함한다. 따라서, 기재된 상기 도메인들의 상기 개시 및 종결 아미노산들은 앞서 기재된 함페 및 왕에 의한 논문들에서 특정된 도메인 영역들과는 어느 정도 다르다.
아비딘 융합 단백질들의 발현
T75 조직 배양 플라스크들 내의 인간 293T 부착 세포들의 일시적인 감염에 의하여 아비딘 융합 단백질들의 발현이 달성되었다. 세포들은 10%의 투석된 FBS 및 1x 펜-스트렙-글루타민(1x Pen-strep-glutamine ; 생장배지(growth medium))를 갖는 DMEM(고농도의 글루코스) 내에서 37℃ 및 5% CO2에서 성장되고 그리고 유지되었다. 거의 3*106 293T 세포들을 15㎖의 생장배지들을 포함하는 T75 플라스크들 내로 접종시키고, 그리고 대략 20시간 동안 밤새도록 성장시켰다. 그 다음에 세포들을 pCEP4-아비딘(N)-c-fms 구축물들로 형질변환시켰다. 각 플라스크 내에, 15㎍의 DNA를 옵티-멤 배지(Opti-MEM medium ; 인비트로겐)의 존재 중에서 75㎕의 리포펙타민 2000(Lipofectamine 2000 ; 인비트로겐)들과 혼합시키고, 그리고 그 착물을 20분간 배양시켰다. 상기 형질변환 착물을 대응하는 플라스크 내로 접종시키고 그리고 옵티-멤 배지 내에서 37℃에서 4 내지 5시간 동안 배양시켰다. 상기 배양 시간의 끝에서, 상기 옵티-멤 배지를 새로운 생장배지로 교체하였다. 형질변환의 대략 48시간 후에, 상기 조정된 배지를 수확하고 그리고 4℃에서 2000*g에서 10분간 원심분리시켜 세포들과 부스러기(debris)들을 제거하고 그리고 50㎖ 시험관들에로 옮겼다. 대조 플라스크는 또한 DNA가 사용되지 않았다는 것(가짜 형질변환)을 제외하고는 동일한 프로토콜을 따라 준비하였으며, 결합 실험들을 위한 음성(negative)의 대조 조건화된 배지를 수득하였다.
융합 단백질들의 검출
각 c-fms 아비딘 융합 단백질의 농도는 정량적 FACS 기반 분석(quantitative FACS based assay)을 사용하여 결정되었다. 상기 c-fms 아비딘 융합 단백질을 6.7㎛ 비오틴 폴리스티렌 비드(biotin polystyrene beads ; 미합중국 일리노이주 리버티빌 소재의 스페로테크 인코포레이티드(Spherotech, Inc.))들 상에 포획하였다. 1배(1X)의 조정된 배지(20 및 200㎕)들을 5㎕(약 3.5*105) 비드들 상에 첨가하고 그리고 실온에서 회전시키면서 1시간 동안 배양시켰다. 조정된 배지를 원심분리에 의해 제거하고 그리고 샘플들을 0.5% BSA를 포함하는 PBS(BPBS)로 세척하였다. 상기 아비딘 비드들을 BPBS 내의 염소 FITC-표지화된 항-아비딘 항체(미합중국 캘리포니아주 벌링검 소재의 벡터 랩스(Vector Labs)) 0.5㎍/㎖ 용액 200㎕로 45분간 실온에서 박막(foil)로 덮어서 염색시켰다. 배양 후, 상기 비드들을 원심분리에 의해 재수집하고, BPBS로 세척하고 그리고 분석을 위하여 0.5㎖ BPBS 내에 재현탁시켰다. FITC 형광을 FACScan(미합중국 뉴저지주 프랭클린 레이크스 소재의 벡톤 딕킨슨 바이오사이언스(Becton Dickinson Bioscience))를 사용하여 검출하였다. 시그널을 r아비딘(rAvidin)에 대해 유도된 표준 곡선을 사용하여 단백질 질량으로 전환시켰다. 에피토프 매핑을 위하여, 비오틴 비드들을 3.5*105 비드들 당 약 100ng의 c-fms 아비딘 융합 단백질로 담지시키고 그리고 생장배지로 용적을 증가시켰다.
항체 결합
FACS
분석
정규화된 양의 c-fms 서브도메인 융합 단백질들로 담지된 비오틴-코팅된 폴리스티렌 비드(스페로테크 인코포레이티드)들을 0.2㎖의 BPBS 내의 1㎍의 FITC 콘쥬게이트된 항 c-fms 모노클로날 항체(1.109, 1.2 및 2.36)와 혼합하였다. 실온에서 1시간 동안의 배양 후, 3㎖의 세척완충제(washing buffer ; BPBS)를 첨가하고 그리고 항체-비드 복합체들을 750*g에서 5분간의 원심분리에 의해 수집하였다. 펠릿을 3㎖의 BPBS로 세척하였다. 아비딘-비드 복합체들 상에 결합된 상기 항체를 FACS(벡톤 딕킨슨) 분석에 의해 검출하였다. 각 샘플에 대하여 평균(X) 형광 세기(mean (X) fluorescent intensity)를 기록하였다.
항체 경쟁 분석
플루오르세인으로의 표지를 준비하기 위하여, 모노클로날 항체들을 투석시키고 그리고 PBS(pH 7.4) 내의 1㎎/㎖의 농도로 재현탁시켰다. 분자프로브(Molecular Probes(F-2181)로부터의 이성질체(isomers)들과 혼합된 표지([6-플루오르세인-5-(및-6)-카르복사미도]헥산온산, 숙신이미딜에스테르5(6)-SFX)를 DMSO 내의 5㎎/㎖의 표지스톡(label stock)으로부터 상기 단백질에 몰비 9.5:1(표지:단백질)로 첨가하였다. 상기 혼합물을 실온에서 암실 내에서 1시간 동안 배양시켰다. PBS 내에서의 투석에 의하여 상기 표지화된 항체를 유리 표지로부터 분리하였다. 각 경쟁 실험들에 대하여, 20-배 과량(20㎍/㎖)의 표지화되지 않은 경쟁 항체, BPBS내의 아비딘 융합 단백질로 코팅된 3*105 비오틴 비드들을 포함하는 결합반응을 수집하였다. 표지화되지 않은 경쟁 항체의 30분의 사전-배양 후에 상기 FITC-표지화된 항체(1㎍/㎖)를 첨가하였다. 상기 과정 후에 이 시점으로부터 앞으로 색채법(color method)을 수행하였다.
293T 세포들 내에서 발현된 상기 융합 단백질들 전부(도 11)가 FACScan에 의해 FITC-표지화 항-아비딘 항체와 함께 검출될 수 있다. 어느 c-fms Ig-형 도메인이 항체-결합 사이트인지를 결정하기 위하여, 6개의 융합 단백질들 전부를 결합분석에서 사용하였다. 상기 항체 클론 1.109, 1.2 및 2.36들이 인간 c-fms 서브도메인 Ig-형 1-2, Ig-형 1-3, Ig-형 l-4 및 Ig-형 1-5 융합 단백질들에 결합한다. 이들은 단일의 도메인 c-fms Ig-형 l 및 Ig-형 2 융합 단백질들에는 결합하지 않는다. 비교를 위하여, 인간 c-fms ECD가 양성의 대조로서 사용되었다(도 11 및 도 12). 이들 결과들은 이들 3개의 항체들이 주로 인간 c-fms의 N-말단의 Ig-형 루프 1 및 Ig-형 루프 2들에 위치하며 상기 Ig-형 루프 1 및 상기 Ig-형 루프 2 영역들 둘 다의 존재를 요구한다는 것을 나타내고 있다. 상기 결과들은 또한 상기 항체가 주로 Ig-형 루프 3에 위치되는 상기 리간드의 높은 친화성의 결합 사이트를 직접적으로 차단하지는 않을 것이라는 것을 나타내고 있다. 이는 둘 달 리간드 결합에 대하여 절대적인 영역들인 Ig-형 루프 1 및 2들로 인하여 상기 리간드 결합에 간접적으로 영향을 미칠 수 있을 것이다(왕과 그의 동료들의 1993, Molecular Cell Biology 13:5348-5359).
3개의 항체들 중, 1㎍/㎖ 하에서 다른 2개의 항체들에 비해 클론 1.109가 가장 높은 결합 시그널을 가졌다. 상기 경쟁 데이터는 상기 항체들 3개가 20배 과량의 표지화되지 않은 항체에 대하여 서로 차단할 수 있다는 것을 입증하고 있다(도 13, 도 14 및 도 15들을 참조하시오). 상기 경쟁 데이터는 또한 이들 3개의 항체들의 에피토프들이 Ig-형 루프 1 및 2와 유사하거나 또는 인접하고 있다는 것을 나타내고 있다.
실시예 13 : 항-c-
fms
항체 1.2
SM
대 통상의 항체들의
에피토프
매핑
이 실험은 본 명세서에서 기술된 상기 인간 항체들 중의 특정의 것이 다수의 상용적으로 획득가능한 항체들과 동일하거나 또는 다른 에피토프에 결합하는 지를 결정하기 위하여 수행되었다.
재료들 및 방법들 :
시험된
상용적인
c-
fms
항체들
시험된 생쥐 및 집쥐 항체들을 표 21 및 표 22들에 나타내었다.
| 원천(source) | 물품번호(Cat. No.) 및 클론 번호 | 속기표기 | 제조업자 마다의 결합 영역 |
| 바이오소스 (Biosource) |
Cat. No. AHT1512, 클론 2-4A5-2 |
2-4A-5-2 | 아미노산 349-512 사이 |
| 유에스 바이올로지컬 (US Biological) |
Cat. No. C2447-53, 클론 5J15 |
5J15 | 아미노산 349-512 사이 |
| 유에스 바이올로지컬 | Cat. No. C2447-50, 클론 O.N. 178 |
0N178 | 기술되지 않음 |
| 원천(source) | 물품번호(Cat. No.) 및 클론 번호 | 속기표기 | 제조업자 마다의 결합 영역 |
| 알앤드디 시스템즈 (R&D Systems) |
Cat. No. MAB329, 클론 61708 |
MAB329 | 면역원으로 사용된 세포외 도메인 |
| 알앤드디 시스템즈 | Cat. No. MAB3291, 클론 61701 |
MAB3291 | 면역원으로 사용된 세포외 도메인 |
| 알앤드디 시스템즈 | Cat. No. MAB3292, 클론 61715 |
MAB3292 | 면역원으로 사용된 세포외 도메인 |
c-fms-아비딘 융합 구축물들 및
아비딘
융합 단백질들의 발현
인간 c-fms 아비딘 융합 단백질 구축물들을 실시예 12에서 기술한 바와 같이 준비하였다. 10㎝ 조직 배양 플레이트들 내의 인간 293T 부착 세포들의 일시적인 감염에 의하여 아비딘 융합 단백질들의 발현이 달성되었다. 세포들은 5%의 정량화된 FBS 및 1배의 펜-스트렙-글루타민(인비트로겐), 1배의 비-필수적인 아미노산(인비트로겐) 및 1배의 피루브산나트륨(인비트로겐)(생장배지)로 보충된 DMEM(고농도의 글루코스) 내에서 37℃ 및 5% CO2에서 성장되고 그리고 유지되었다. 거의 2.5*106 293T 세포들을 10㎖의 생장배지들을 포함하는 10㎝ 플레이트들 내로 접종시키고, 그리고 대략 20시간 동안 밤새도록 성장시켰다. 그 다음에 세포들을 pCEP4-아비딘(N)-c-fms 구축물들로 형질변환시켰다. 각 형질변환들에 대하여, 7.5㎍의 DNA를 무보충의 DMEM 배지(인비트로겐)의 존재 중에서 45㎕의 푸젠6(FuGene6 ; 로쉬(Roche)와 혼합시키고, 그리고 그 착물을 20분간 배양시켰다. 상기 형질변환 착물을 대응하는 플레이트에 첨가하고 그리고 37℃에서 밤새도록 배양시켰다. 다음날 아침, 상기 세포들을 1배의 둘베코의 인산염 완충된 식염수(Dulbecco's Phosphate Buffered Saline ; PBS)(인비트로겐)으로 세척하고 그리고 앞서 언급한 보충물들 더하기 인슐린(Insulin), 트랜스페린(Transferrin), 셀레늄-X(Selenium-X ; ITS-X)(인비트로겐)들을 포함하는 무혈청의 DMEM 5㎖를 공급하였다. 형질변환의 대략 48시간 후에, 상기 조정된 배지를 수확하고 그리고 4℃에서 2000*g에서 10분간 원심분리시켜 세포들과 부스러기(debris)들을 제거하고 그리고 15㎖ 시험관들에로 옮겼다. 대조 플레이트는 또한 DNA가 사용되지 않았다는 것(가짜 형질변환)을 제외하고는 동일한 프로토콜을 따라 준비하였으며, 결합 실험들을 위한 음성의 대조 조건화된 배지를 수득하였다.
융합 단백질들의 검출
각 c-fms 아비딘 융합 단백질의 농도는 정량적 FACS 기반 분석을 사용하여 결정되었다. 아비딘 융합 단백질을 6.7㎛ 비오틴 폴리스티렌 비드(미합중국 일리노이주 리버티빌 소재의 스페로테크 인코포레이티드)들 상에 포획하였다. 1배의 조정된 배지(2, 20 및 200㎕)들을 5㎕(약 3.5*105) 비드들 상에 첨가하고 그리고 실온에서 회전시키면서 1시간 동안 배양시켰다. 조정된 배지를 원심분리에 의해 제거하고 그리고 샘플들을 2% FBS를 포함하는 PBS(FPBS)로 세척하였다. 상기 아비딘 비드들을 FPBS 내의 FITC-표지화된 염소 항-아비딘 항체(미합중국 캘리포니아주 벌링검 소재의 벡터 랩스) 1.0㎍/㎖ 용액 500㎕로 30분간 실온에서 회전시키면서 염색시켰다. 배양 후, 상기 비드들을 원심분리에 의해 재수집하고, FPBS로 세척하고 그리고 분석을 위하여 0.5㎖ FPBS 내에 재현탁시켰다. FITC 형광을 FACScan(미합중국 뉴저지주 프랭클린 레이크스 소재의 벡톤 딕킨슨 바이오사이언스)를 사용하여 검출하였다. 시그널을 r아비딘에 대해 유도된 표준 곡선을 사용하여 단백질 질량으로 전환시켰다. 에피토프 매핑을 위하여, 비오틴 비드들을 3.5*105 비드들 당 약 200ng의 c-fms 아비딘 융합 단백질로 담지시키고 그리고 FPBS로 용적을 증가시켰다.
항체 결합
FACS
분석
정규화된 양의 c-fms 서브도메인 융합 단백질들로 담지된 비오틴-코팅된 폴리스티렌 비드(스페로테크 인코포레이티드)들을 0.2㎖의 FPBS 내의 1㎍의 인간 항 c-fms 모노클로날 항체(1.2) 생쥐 항 c-fms 모노클로날 항체(MAB 329, MAB 3291 및 MAB3292 [알앤드디 시스템즈(R&D Systems)]) 또는 생쥐 항-c-fms 모노클로날 항체(2-4A5-2 [인비트로겐], O.N.178 및 5J15 [유에스바이올로지컬(U.S. Biological)])과 혼합하였다. 실온에서 1시간 동안의 배양 후, 상기 항체-비드 복합체들을 1.25㎖의 세척완충제(FPBS)로 세척하고 세척들 사이에서 18,000*g에서 1분간의 원심분리에 의한 수집을 병행하였다. 계속해서 상기 항체들을 1.0㎍/㎖에서 30분간 FITC(사우던 바이오테크(Southern Biotech))에 콘쥬게이트된 종들에 적절한 염소 2차 항체로 염색시켰다. 상기 세척단계들을 반복하고 그리고 분석을 위하여 상기 항체-비드 복합체들을 0.5㎖ FPBS 내에 재현탁시켰다. 아비딘-비드 복합체들 상에 결합된 상기 항체를 FACS(벡톤 딕킨슨) 분석에 의해 검출하였다. 각 샘플에 대하여 평균(X) 형광 세기를 기록하였다.
항체 경쟁 분석
플루오르세인으로의 표지를 준비하기 위하여, 모노클로날 항체들을 투석시키고 그리고 PBS(pH 7.4) 내의 1㎎/㎖의 농도로 재현탁시켰다. 분자프로브(F-2181)로부터의 이성질체(isomers)들과 혼합된 표지([6-플루오르세인-5-(및-6)-카르복사미도]헥산온산, 숙신이미딜에스테르5(6)-SFX)를 DMSO 내의 10㎎/㎖의 표지스톡으로부터 상기 단백질에 몰비 10:1(표지:단백질)로 첨가하였다. 상기 혼합물을 실온에서 암실 내에서 1시간 동안 배양시켰다. PBS 내에서의 NAP 5 컬럼 크로마토그래피 및 후속하는 0.2㎛ 여과에 의하여 상기 표지화된 항체를 유리 표지로부터 분리하였다. 각 경쟁 실험들에 대하여, 25-배 과량(25㎍/㎖)의 표지화되지 않은 경쟁 항체, FPBS내의 아비딘 융합 단백질로 코팅된 3.5*105 비오틴 비드들을 포함하는 결합반응을 수집하였다. 15분의 사전-배양 후에 상기 FITC-표지화된 항체(1㎍/㎖)를 첨가하였다. 상기 과정 후에 이 시점으로부터 앞으로 색채법을 수행하였다.
결과들 및 논의
293T 세포들 내에서 발현된 상기 융합 단백질들 전부가 FACScan에 의해 FITC-표지화 항-아비딘 항체와 함께 검출될 수 있다. 실시예 12에서 기술한 바와 같이, 시험된 여러 항체들이 Ig1-2 아비딘 융합 구축물 내에서 발견되는 Ig-형 루프 1 및 Ig-형 루프 2 영역들 둘 다의 존재를 필요로 하는 유사한 에피토프들에 결합하였다. 따라서, 본 명세서에서 제공되는 인간 항체들, 상용적으로 획득가능한 항-인간 c-fms 항체들 및 아비딘 융합 구축물들의 패널의 선택 구성(select members)들 중의 하나로 결합 및 경쟁 실험들이 수행되었다.
기대된 바와 같이 상기 상용적인 항체들 전부가 성공적으로 전장의 c-fms ECD Ig1-5 구축물들에 결합할 수 있었다. 상기 6개의 상용적인 항체들 중에서, 하나(MAB 3291)은 상기 Ig1-2 구축물에 결합할 수 있었으며, 상기 인간 항-c-fms 에피토프들에 대한 가능한 경쟁자(competitor)를 나타내었다. 다른 결합 실험들이 상기 Ig1 구축물을 사용하여 수행되었다. MAB3291은 상기 Ig1 구축물을 결합하는 것으로 나타났으며, 그의 에피토프가 전적으로 상기 Ig1-형 도메인 내에 위치됨을 나타내었다. 상기 Ig1 및 Ig1-2 구축물들 내의 MAB329에 대해 보여진 약간의 시그널은 상기 비드들에 대한 상기 항체의 배경결합(background binding)임을 확인하였다.
상기 경쟁 데이터는 상기 상용적인 출처의 항체들 중 어느 것도 심지어 25배의 과량의 경쟁자 항체에서 대표적인 인간 항체를 차단할 수 없다는 것을 입증하고 있다.
상기 결합 및 경쟁 실험들로부터의 결합된 데이터는 상기 상용적인 항체들이 상기 인간 항-c-fms 항체들에 의해 활용되지 않는 에피토프들에 결합한다는 것을 입증하고 있다.
실시예
14 : c-
fms
의 아르기닌/
글루타민산
스캐닝에 의한 항 c-
fms
항체들의 에피토프
매핑
c-fms에 대한 항체 결합을 매핑하는 데 아르기닌/글루타민산-스캐닝 전략(arginine/glutamic acid-scanning strategy)이 사용되었다. 상기 아르기닌 및 글루타민산 측쇄들은 하전되고 그리고 벌키(bulky ; 부피가 큰)하며, 또한 c-fms에 대한 항체 결합을 방해할 수 있다. 따라서 이 방법은 음성적으로 돌연변이되는 경우에 잔기들이 상기 항원 결합 단백질의 c-fms에의 결합에 영향을 줄 수 있다는 것을 나타낼 수 있다. 이는 돌연변이되지 않은 항원 결합 단백질 내의 잔기들이 상기 항원 결합 단백질과 접촉하거나 또는 상기 항체에 긴밀하게 근접하여 아르기닌 또는 글루타민산으로의 치환이 결합에 영향을 주기에 충분하도록 한다는 것을 나타내고 있다.
아르기닌/
글루타민산
돌연변이들의 구축, 발현 및 특정화
c-fms의 상기 제1의 3개의 Ig 도메인들 전체를 통하여 분포되는 95개의 아미노산들이 돌연변이를 위하여 선택되었다. 상기 선택은 잘못 중첩된 단백질(misfolded protein)의 결과를 가져오는 돌연변이의 가능성을 감소시키기 위하여 시스테인 및 프롤린을 제외하고는 하전되거나 또는 극성의 아미노산들로 편위되어 있다. 비-아르기닌 아미노산들을 아르기닌으로 돌연변이시켰으며; 아르기닌 및 리신들은 글루타민산으로 돌연변이시켰다.
상기 돌연변이된 잔기들을 포함하는 감작 및 비-감작의 올리고뉴클레오티드들을 96-웰 포맷(96-well format) 내에서 합성시켰다. 상기 c-fms 세포외 도메인-Flag-His-태그된 구축물("야생형")의 돌연변이생성을 퀵체인지 II 키트(Quikchange II kit ; 스트라타젠(Stratagene), #200523)를 사용하여 수행하였다. pTT5 벡터 내의 Flag-His-태그된 c-fms의 모든 돌연변이 구축물들을 96-웰 플레이트들 내의 일시적으로 형질변환된 293-6E 현탁 세포들(NRCC) 내에서 발현시켰다. 조정된 배지 내에서의 재조합 단백질의 발현 수준(expression levels)들 및 완전성(integrity)은 His-태그에 대한 웨스턴블럿 및 후속하는 항-아이소타입 알렉사-플루오르 항체(anti-isotype Alexa-fluor antibody)에 의해 특정화되었다. 후속하는 에피토프 매핑 실험들이 조정된 배지 내의 단백질을 사용하여 수행되었다.
돌연변이 발현은 이페이지 (ePage) SDS-PAGE 전기천공 장치(인비트로겐) 상에서의 각 웰로부터의 상청액들의 런닝, 블럿팅 및 항-His 항체(노바젠(Novagen))로의 탐침 및 후속하는 항-아이소타입 알렉사-플루오르 항체에 의해 특정화되었다. 각 돌연변이 구축물은 발현되었다.
형태학적
에피토프들의
결정
항-c-fms 항체들이 c-fms 상의 형태학적 에피토프에 결합하는 지의 여부를 결정하기 위하여, 3개의 항-c-fms 항체들(1.2 SM, 1.109 SM 및 2.360) 및 c-fms들을 개별적으로 환원 및 비-환원 조건들(reducing and non-reducing conditions) 하에서 웨스턴블럿들에 대하여 런닝시켰다. 환원조건들 하에서의 웨스턴블럿들에서의 결합들의 결여에 의해 입증된 바와 같이 항체 1.2 SM 및 2.360들은 형태학적 에피토프에 결합하는 것을 나타내었으며, 반면에 항체 1.109 SM에 대하여 가벼운 밴드(light band)가 관측되어 이것이 선형의 에피토프에 결합할 수 있음을 나타내었다.
바이오플렉스
결합 분석(
BioPlex
binding
assay
)
비드-기반 멀티플렉스 분석(bead-based multiplexed assay)을 사용하여 상기 95개의 c-fms 돌연변이들, 야생형 및 음성의 대조들에의 항체 결합을 동시적으로 측정하였다. 100개의 세트들의 컬러-코드화된 스트레파비딘-코팅된 룸아비딘 비드(color-coded strepavidin-coated LumAvidin beads ; 루미넥스(Luminex))들을 비오티닐화된 항-pentaHis 항체(키아젠, #1019225)에 대하여 1시간 동안 실온(RT)에서 결합시키고 계속해서 세척하였다. 계속해서 각 컬러-코드화된 비드 세트를 100㎕ 상청액 내에서 1시간 동안 실온에서 c-fms 돌연변이, 야생형 또는 음성의 대조에 결합하도록 하였으며, 세척하였다.
각각이 특정의 단백질과 관련된 상기 컬러-코드화된 비드 세트들을 수집하였다. 상기 수집된 비드들을 96-웰 필터 플레이트(96-well filter plate ; 밀리포어(Millipore), #MSBVN1250)의 96개의 웰들에 분획시켰다. 3중의 지점(triplicate points)들을 위한 3배 희석의 100㎕의 항-c-fms 항체(1.2 SM, 1.109 SM 및 2.360)들을 3개의 컬럼들에 첨가하고 그리고 실온에서 1시간 동안 배양시킨 후, 세척하였다. 1:200 희석 파이코에리트린(PE)-콘쥬게이트된 항-인간 IgG Fc(잭슨 임뮤노리서치(Jackson Immunoresearch), #109-116-170) 100㎕를 각 웰에 첨가하고 그리고 실온에서 1시간 동안 배양시킨 후, 세척하였다.
비드들을 PBS 내의 1% BSA에 재현탁시키고, 10분간 진탕시키고 그리고 바이오플렉스 기구(바이오-라드(Bio-Rad)) 상에서 판독하였다. 상기 기구는 각 비드를 그의 컬러-코드로 동정하고 그리고 상기 PE 염료의 형광 세기에 따라 상기 비드들에 결합된 항체의 양을 측정하였다. 각 돌연변이에의 항체 결합을 직접적으로 동일한 풀(pool) 내에서의 야생형에 대한 그의 결합을 직접적으로 비교하였다.
돌연변이 c-
fms
에의 항체 결합의 동정
상기 분석 시스템의 변동성(variability) 및 결합에서의 변화들의 명료성(significance)들을 경험적으로 결정하였다. 야생형 c-fms의 100세트 전부의 컬러-코드화된 비드들에의 결합에 의해 비드-대-비드 및 웰-대-웰 변동성을 실험적으로 결정하였다. 비드들을 96-웰 플레이트의 각 웰에 분배하고 그리고 상기 플레이트의 각 컬럼에 상기 플레이트의 12개의 컬럼들 전부를 가로질러 3배 희석들의 항-c-fms 항체 1.2 SM으로 탐침시켰다. 최대 시그널들, 최소 시그널들 및 경사의 다양성을 측정하는 것으로부터의 곡선 피트(curve fits)들을 사용하여 EC50을 파생시켰다. 다양성 측정들을 사용하여 EC50들에서의 등급 이동(magnitude shift)이 명확한 지의 여부를 결정하였다.
가중된 4 변수 로지스틱 커브 피트(weighted 4 Parameter Logistical curve fit ; Splus 내의 VarPower를 갖는 4PL)를 이용하여 항체 결합의 평균형광세기(MFI ; mean fluorescence intensity)를 그래프로 그렸다. 각 풀 내의 3개의 야생형 대조들을 사용하여 실험적 변동성을 결정하였다. 돌연변이 항원에의 항체 결합을 각 야생형 대조와 비교하였다. 돌연변이와 각 야생형 대조 사이의 EC50 중첩 변화의 99% 신뢰구간(CI ; confidence interval)을 산출하였으며, 보다 큰 p-값을 주는 야생형 대조에의 비교가 보고되었다. 벤자미니-호쉬베르그 오류발견율(Benjamini-Hochberg False Discovery Rate ; FDR) 대조를 사용하는 다중도 조정(multiplicity adjustment)이 적용되었다. 0.02% 이하의 p-값 조정된 FDR을 갖는, 그의 EC50의 99% CI가 야생형 EC50과 명백하게 다른 돌연변이들이 상기 단백질 항원과 항원 결합 단백질 사이의 특이적인 결합 반응에서 중요한 것으로 고려되었다. 게다가, 야생형의 30% 또는 그 이하로의 최대 MFI 시그널에서의 감소에 의해 입증된 바와 같이 결합이 감소된 돌연변이들이 상기 단백질 항원과 항원 결합 단백질 간의 결합에 명백하게 영향을 주는 것으로 고려되었다. 표 23(항체 결합에 영향을 주는 돌연변이들의 요약)은 인간 c-fms의 상기 세포외 도메인에 결합하는 1.2 SM, 1.109 및 2.36 항체들의 능력을 명백하게 감소시키는 돌연변이들의 "히트(hits)" 또는 위치를 요약하고 있다. 표 23에서 사용된 표기법은 (야생형 잔기 : 폴리펩티드 내의 위치 : 돌연변이 잔기)이며, 여기에서 번호들은 SEQ ID NO: 1에서 나타낸 바와 같다.
[표 23]
적어도 하나의 항체의 결합이 표 23에 나타낸 특정의 돌연변이들의 존재 중에서 유지되기 때문에, 상기 돌연변이 단백질들은 도입된 돌연변이로 인하여 극도로 잘못 중첩되거나 또는 응집될 것으로는 여겨지지 않는다. 이는 또한 상기 항체들이 여전히 항원에 결합할 수 있기 때문에 상기 항체들의 전부에 대해 EC50 시프트(shift)를 야기하는 돌연변이들에 대해서도 사실이다. 비록 시험된 항체들 각각이 분류 분석(binning analysis)에 의하여 나타난 바와 같은 유사한 영역에 결합함에도 불구하고, 각 항체는 항체가 돌연변이 항원에 결합하는 것을 저해하는 돌연변이들에 의해 구별될 수 있을 것이다. 상기 돌연변이들의 일부가 다중의 항체들에 영향을 준다는 것은 상기 항체들이 유사한 분류(similar bins)들에 속한다는 사실과 일치한다.
실시예
15 :
MDAMB231
유방
선암
이종이식의 성장의 저해
본 명세서에서 제공되는 상기 항원 결합 단백질들이 생쥐 c-fms가 아닌 인간 c-fms에 결합하기 때문에, 쥣과의 동물의 c-fms에 결합하는 항체들에 대하여 일련의 생체 내 실험들을 수행하여 암을 치료하기 위한 항-c-fms 항체의 유용성을 증명하였다.
무흉선의 누드생쥐(athymic nude mice)들에 매트리겔(Matrigel)의 존재(1 : 1) 중에서의 1,000만(10 million)의 MDAMB231 인간 유방 선암 세포들을 피하로 이식하였다. 종양 세포 이식 1일 이내에 시작하여, 생쥐들에 100㎕의 PBS 내의 400㎍의 항-쥣과의 동물의 c-fms 또는 400㎍의 대조 생쥐 항-생쥐 IgG들을 연구의 기간 동안에 주당 3회씩 복강내로 주사하였다. 종양 측정 및 치료 일수들을 도 16에 나타내었다. 51일 후, 생쥐들을 안락사시키고, 그리고 종양들을 수집하여 포르말린 고정시켰다. H&E 염색된 부위들 및 F4/80(마크로파지 마커) 표적화된 면역조직화학법(immunohistochemistry) 부위들을 평가하였다. 맹검그룹과 처치그룹에 대해 모든 스코어링(scoring)이 수행되었다. 항-쥣과의 동물의 c-fms 항체로 처치된 생쥐로부터의 부위들이 대조로 처리된 생쥐들 보다 명백하게 낮은 염색을 나타내었으며, 따라서 종양 연관 마크로파지들의 수에서의 명백한 감소를 나타내었다. 괴사의 정도를 보다 객관적으로 평가하기 위하여, AFOG 염색된 부위들의 디지털 이미지들을 메타부 소프트웨어(Metavue software)를 사용하여 캡쳐하였으며, 종양들의 전체 단면영역 및 괴사 단면영역들을 측정하였다. 계속해서 이들 측정들로부터 각 종양의 괴사 백분율을 산출하였으며, 도 16에 나타내었다. 이들 결과들은 항-c-fms 항체가 종양 연관 마크로파지들을 감소시키고, 괴사를 증가시키고 그리고 MDAMB231 유방 선암 이종이식들의 성장을 저해할 수 있다는 것을 입증하고 있다.
실시예 16 : 구축된 NCIHl975 폐 선암 이종이식의 성장의 저해
무흉선의 누드생쥐들에 매트리겔의 존재(1 : 1) 중에서의 1,000만(10 million)의 NCIHl975 폐 선암 세포들을 피하로 이식하였다. 종양들이 250 내지 300㎣까지 성장하도록 한 후, 생쥐들에 100㎕의 PBS 내의 400㎍의 항-쥣과의 동물의 c-fms 또는 400㎍의 대조 집쥐 항-생쥐 IgG들을 연구의 기간 동안에 주당 3회씩 복강내로 주사하였다. 생쥐들의 제3의 그룹을 30㎎/㎏의 탁소테르(Taxotere)(양성 대조)로 주당 1회씩 주사하였다. 종양 측정 및 치료 일수들을 도 17에 나타내었으며, 이는 항-c-fms 항체가 구축된 NCIHl975 폐 선암 이종이식의 성장을 저해할 수 있다는 것을 나타내고 있다.
상기 종양 모델들은 본 명세서에서 기술되는 바와 같이 암의 치료에 이용될 수 있는 상기 CSF-1/cfms 축의 활성을 저해하는 항-cfms 항체의 유용성을 입증하고 있다. 병 든 조직 내에서의 마크로파지들의 침입을 감소시키는 이러한 항체들의 능력은 상기 항체들이 또한 대사성 및 염증성 질병들에 사용될 수 있다는 것을 의미한다.
실시예
17 : 신생혈관생성을 제어하기 위한 상기
CSF
-1/
CSF
-1R의 모듈화
마크로파지들의 CSF-1 매개 회복(CSF-I mediated recruitment), 분화 및 자극들이 종양들 및 다른 정상적인 조직들 내에서의 신생혈관생성에 관여하는 지의 여부를 시험하기 위하여, 2개의 서로 다른 중화 생쥐 항-쥣과의 동물의 CSF-1R 모노클로날 항체들(M279는 내부적으로 생성된 것이고; 다른 하나인 FS98은 에비오사이언시즈(Ebiosciences)로부터 수득된 것임)을 생체 내에서 생쥐 각막의 신생혈관생성에 대한 그들의 효과에 대하여 평가하였다. 대조 mAb, M279 또는AFS98의 단일의 전신적인 투여량 5일 후, 하기 변수들, 즉 1) 상기 생쥐 각막의 신생혈관생성 응답에 연관되는 혈관의 밀도(vascular density), 2) 생쥐 CSF-1의 순환 수준들 및 3) 상기 각막 및 다른 조직들 내로의 마크로파지의 침입의 수준들을 측정하고 그리고 분석하였다.
생쥐 각막 포켓 분석(
Mouse
Corneal
Pocket
Assay
)
4㎜의 PVC 스펀지(엠-팩트 국제(M-PACT Worldwide), 유도라(Eudora), KS)를 정밀하게 2개의 동일한 조각들로 절단하고 그리고 2.4㎍의 재조합 인간 FGF-2 또는 48㎍의 VEGF(미합중국 미네소타주 미니애폴리스 소재의 알앤디 시스템즈(R&D Systems))를 포함하는 8㎕의 PBS 내에 함침시켰다. 계속해서 상기 스펀지를 각막 포켓 이식을 위하여 적절한 48개의 유사한 크기의 미니-스펀지 단편들(펠릿들)로 무균적으로 가공하였다. 각 스펀지 단편은 대략 50ng의 재조합 인간 FGF-2 또는 1㎍의 재조합 인간 VEGF를 포함하였다. 암컷 C57 BL/6 생쥐(7 내지 10주령)들을 전신마취를 사용하여 마취시켰으며, 각 안구에 한 방울의 프로파라카인 국소 마취제(Proparacaine topical anesthetic)를 투입하는 것에 의하여 각막 절개를 위한 안구들을 준비하였다. 상기 각막의 중앙 내에 미세한 슬릿을 생성시키고, 그리고 가장자리로부터 대략 1㎜로 안구의 곡면을 따르는 각막간질(corneal stroma) 내에 개구부(opening)("포켓")를 형성시켰다. PBS, VEGF 또는 FGF-2를 포함하는 펠릿들을 상기 각막의 포켓들 내로 위치시키고, 그리고 동물들을 200㎕ 무발열원(pyrogen-free)의 PBS 내의 250㎍의 투여량의 집쥐 IgG(미합중국 미주리주 세인트루이스 소재의 시그마(Sigma)) IP로 또는 250㎍의 정제된 집쥐 항-생쥐 CSF-1R로처리하였다. 펠릿 이식 5일 후에, 생쥐들을 전신 마취로 마취시켰으며, 각 안구(각막)를 상기 펠릿을 포함하는 정중선(meridian) 내에서 극축(polar axis)로부터 45°의 초기각(incipient angle)에 근수직 일루미네이터(near vertical illuminator)와 함께 인사이트 스폿 디지털 카메라(Insight Spot digital camera)가 장착된 입체현미경 하에서 이미지화하였다. 이들 획득된 디지털 이미지들을 감색필터(subtractive color filter ; 어도비 포토샵(Adobe Photoshop))을 사용하여 처리하였으며, 바이오퀀트 이미지 분석 소프트웨어(Bioquant image analysis software ; 미합중국 테네시주 내쉬빌(Nashville))를 사용하여 분석하여 문턱 매칭 가시 모세관(threshold matching visible capillaries)들을 초과하는 각막 주변의 총 밀도 내의 화소(pixels)들의 분수(fraction)를 결정하였다. 상기 각막의 총 혈관밀도를 화소들의 분수를 사용하여 결정하였으며, 그 결과를 전체 안구면적 화소수에 대한 혈관 면적 화소수의 비로 표현하였다.
생쥐
CSF
-1 효소면역측정법
생쥐 CSF-1의 혈청 수준들을 제조업자의 지시들에 따라 듀오셋 항체 효소면역측정법 시스템(DUOSET antibody ELISA system ; 알앤디 시스템즈)를 사용하여 항 CSF-1R 항체 활성의 바이오마커(biomarker)로서 결정하였다.
생쥐의 간 및 각막 조직들 내의
마크로파지
및 혈관들의 면역위치지정(
Immunolocalization
of
macrophage
and
blood
vessels
in
liver
and
corneal
tissues
of
mice
)
조직 내 마크로파지 및 혈관 수준들에 대한 항-생쥐 CSF-1R 항체들의 영향들을 결정하기 위하여, 알렉사 488(Alexa 488), 클론 BM8(1 : 1000)으로 콘쥬게이트된 집쥐 항-생쥐 F4/80(마크로파지-제한 세포 표면 당단백질)을 사용하여 조직 마크로파지들을 검출하였다. PE로 콘쥬게이트된 CD31, 집쥐 항-생쥐 PECAM-1 IgG2a, 클론 390(1㎍/㎖ 사용)을 사용하여 내피세포(endothelial cells)들을 검출하였다. 조직을 수확한 후, 이를 후속하는 가공을 위하여 OCT 내에서 동결시켰다. 간 또는 각막의 5미크론(micron) 절취부(sections)들을 냉아세톤(cold acetone)으로 실온에서 15분간 고정시켰으며, 계속해서 PBS로 2회 세척하였다. 세척 후, 절취부들을 블록킹용액(blocking solution ; BS) 내에서 30분간 실온에서 배양시켰다. F4/80-488 및 CD31-PE들 둘 다를 앞서의 농도들에서의 BS 내에 첨가하고 그리고 절취부들을 30분간 실온에서 배양시킨 후, 후속하여 PBS로 2회 세척하였다. 슬라이드들을 탑재배지(mounting media) 내에 탑재하고 그리고 라이카-하마무츠-오픈랩 시스템(Leica-Hamamutsu-Openlab system)으로 형광 이미지들을 획득하였다.
결과들 및 논의:
집쥐 항-muCSF-lR 중화 항체들, M279 및 AFS98들은 명백하게 FGF-2를 저해하나, VEGF-유도 생쥐 각막 신생혈관생성은 대략 80%(P<0.01)로 저해하지 못하였다. 단일 투여량 250㎍ M279 또는 AFS98들은 집쥐 IgG-처리 생쥐(45 내지 83배 증가)에서 관측된 수준들과 비교하여 명백하게 muCSF-1 혈청 수준들을 증가시켰다. 생쥐 각막 영역들 내에서의 면역형광 염색/편재화(immunofluorescent staining/localization ; IMF) 결과들은 FGF-2 및 VEGF 펠릿 이식이 외과/PBS 펠릿 이식 단독에 비하여 상기 각막 내에서의 마크로파지 침입을 증가시킨다는 것을 나타내었다. M279 처치는 대조 집쥐 IgG 처치에 비해 대략 85 내지 96%로 자극-유도(FGF-2 및VEGF 둘 다) 각막 마크로파지 침입을 크게 감소시켰다. 생쥐 간 절취부들 내의 IMF 결과들은 또한 M279 또는 AFS98로의 단독의 처치가 명백하게 F4/80 양성 마크로파지들의 수를 상기 생쥐 간 내에서 대략 60%로 감소시킨 반면에 CD31 IMF에 의해 평가된 바와 같이 혈관 밀도를 감지할 만큼 변화시키지는 못하였다(P<0.01). 마크로파지들은 혈관망을 추종하나 대체로 맥관화된 생쥐 각막(vascularized mouse cornea) 내에서의 모세혈관들에 대하여는 상호국한(co-localize) 하지 않는다.
두 신생혈관생성 자극(FGF-2 및 VEGF)들을 평가하는 경우, CSF-1/CSF-1R 상호작용을 차단하는 것은 상기 조직 내로의 마크로파지 침입을 감소시키는 한편으로 이는 단지 각막 혈관밀도 이미징에 기초하여 FGF-2 신생혈관생성을 저해하였다. 이들 결과들에 기초하여, 상기 염증성 환경은 CSF-1 응답 조직 마크로파지들이 신생혈관생성을 용이하게 하거나/촉진하는 경우에 영향을 주는 한편으로 동시에 다중의 염증성 사이트들에서의 상기 조직 마크로파지들이 CSF-1/CSF-1R 상호작용의 진행에 요구되어 상기 염증성 병소에서의 그들의 존재를 유지하도록 한다는 것을 나타내고 있다. 상기 결과들은 염증성 신생혈관생성이 상기 CSF-1/CSF-1R을 저해하는 원발적으로 FGF-2에 의해 구동되는 경우들에서 특히 VEFG 수준들이 높지는 않으나 종양 혈관밀도는 높은 종양들 내에서 새로운 혈관 형성의 감소에 유리할 수 있다는 것을 나타내고 있다.
실시예
18 :
사이노몰거스
원숭이들에서의 독성학
1.2 SM 항체를 사이노몰거스 원숭이들에 투여하였으며, 약물동력학적 마커들을 측정하였다. c-fms 항원 결합 단백질의 영향을 연구하는 데 사용된 코호트를 표 24(독성학 연구를 위한 코호트)에 나타내었다. 항체 1.2 SM을 4주간 주단위로 사이노몰거스 원숭이들에 정맥내 주사에 의해 투여하였으며, 11주의 회복기간이 후속되었으며, 29일차에서 종점 부검(terminal necropsy) 및 3개월에서의 회복 부검(recovery necropsy)을 수행하였다. 혈청 CSF-1 수준들, 타르타르산염-저향성 산포스파타아제 5b(acid phosphatase 5b ; Trap5b), 콜론 마크로파지들의 농도들 및 양들을 포함하는 약물동력학적 마커들이 측정되었다. 이하에서 보다 상세하게 기술되는 바와 같이, 이들 마커들 각각의 측정은 c-fms에 결합하고, 그리고 상기 c-fms/CSF-1 축을 저해하는 상기 항체의 능력을 입증하였다. 상기 마커들의 수준은 또한 혈액 내의 항체의 수준과 연관되어 있다.
| 그룹 | 투여량 (㎎/㎏) |
수컷/암컷의 수 | 수컷/암컷 종점 부검 | 수컷/암컷 회복 부검 |
| 1 | 0 | 5/5 | 3/3 | 2/2 |
| 2 | 20 | 5/5 | 3/3 | 2/2 |
| 3 | 100 | 5/5 | 3/3 | 2/2 |
| 4 | 300 | 5/5 | 3/3 | 2/2 |
항체 1.2
SM
으로의 처치에 대한 혈청
CSF
-1 수준들의 응답
혈청 CSF-1 수준들은 항-c-fms 항체의 존재 및 활성에 대한 바이오마커를 제공한다. 이는 생쥐 내에서의 125I-표지화된 CSF-1의 마크로파지에 의한 선택적 분해(투신스키 알제이(Tushinski RJ)와 그의 동료들의 Cell (1982) 28:71-81); CSF-1이 상기 c-fms 넉아웃 생쥐(knock out mice)들의 혈청 내에서 상승되었다는 관측들(다이 엑스엠(Dai XM)과 그의 동료들의 Blood (2002) 99:111-120); 및 혈청 CSF-1 수준들이 항-생쥐 c-fms 항체로 처치된 생쥐들에서 상승되었다는 입증들에 의하여 입증되었다.
사이노몰거스 CSF-1의 상대적인 농도들이 -7, 8, 29, 57, 85 및 99이들에서 수집된 혈청 시료들에 대하여 결정되었다. 분석법 제조업자들(알앤디 시스템즈 인간 CSF-1 듀오셋 ELISA 키트; 미합중국 미네소타주 미니애폴리스)에 의해 제공된 프로토콜에 따라 효소-연결 면역흡수분석법(ELISA)을 사용하여 샘플들을 분석하였다. 인간 CSF-1 표준 곡선과의 비교에 의하여 사이노몰거스 CSF-1 농도들을 결정하였다.
혈청 항체 1.2 SM의 농도를 측정하기 위하여, 생쥐 항-1.2 SM 항체를 맥시소프 마이크로플레이트 웰들(Maxisorp microplate wells ; Nunc)에 수동적으로 흡수시켰다. 과량의 생쥐 항-1.2 SM 항체를 제거한 후, 상기 마이크로플레이트 웰들을 슈퍼블록®T20(SuperBlock®T20 ; 미합중국 일리노이주 록포드 소재의 피어스(Pierce))로 차단시켰다. 항체 1.2 SM을 100% 사이노몰거스 원숭이 혈청 풀(pool) 내로 스파이킹(spiking)시키는 것에 의하여 표준들 및 품질관리(Standards and quality controls(QCs)들을 준비하였다. 상기 마이크로플레이트 웰들을 블록킹 후에 세척하였다. 상기 표준들, 매트릭스 블랭크(NSB), QCs 및 연구 샘플들을 주변온도로 해동시켰으며, 계속해서 슈퍼블록®T20으로 1/50 전처리한 후 상기 마이크로플레이트 웰들 내로 실었다. 상기 샘플들 내의 상기 항체 1.2 SM을 부동태화된 생쥐 항-1.2 SM 항체로 포획하였다. 상기 마이크로플레이트 웰들을 세척하는 것에 의하여 미결합된 항체 1.2 SM을 제거하였다. 세척 후, 제2의 고추냉이과산화효소(HRP) 콘쥬게이트된 생쥐 항-1.2 SM 항체를 상기 마이크로플레이트 웰들에 첨가하여 상기 포획된 항체 1.2 SM을 결합시켰다. 미결합된 HRP 콘쥬게이트된 항체를 세척에 의하여 제거하였다. 2-성분의 3,3',5,5'-테트라메틸벤지딘(TMB) 기질 용액(2-component 3,3',5,5'-tetramethylbenzidine substrate solution)을 상기 웰들에 첨가하였다. 상기 TMB 기질 용액은 과산화효소와 반응하고, 그리고 고추냉이과산화효소의 존재 중에서 초기 단계에서 캡쳐 시약(capture reagent)에 의해 결합된 항체 1.2 SM의 양에 비례하여 비색 시그널(colorimetric signal)이 생성되었다. 발색현상(color development)을 중단시키고 그리고 상기 색상의 강도(광학적 밀도, OD)를 450 내지 650㎚에서 측정하였다. 1/Y의 가중인자(weighting factor)로 로지스틱(오토-에스티메이트)(4개의 인자 로지스틱; 4-PL)을 사용하는 왓슨 버전 7.0.0.01 리덕션 패키지(Watson version 7.0.0.01 reduction package)를 사용하여 데이터를 환원시켰다.
원숭이들의 항체 1.2 SM으로의 처치는 혈청 CSF-1 수준들에서의 명백한 증가의 결과를 가져왔으며, 이는 항체 혈청 농도와 연관되어 있다. 따라서, CSF-1 혈청 수준들은 항체의 투여 후 및 혈청에서의 그의 축적 후에서 증가하였으며, 계속해서 투여가 완료된 후에는 감소하는 것으로 밝혀졌으며, 상기 혈청 항체 수준들은 감소되었다. 이들 관측들은 상기 c-fms/CSF-1 축에 대해 작용하는 상기 항체와 일치하고 있다.
항체 1.2 SM으로의 처치에 대한 Trap5b 수준들의 응답
Trap5b 수준들은 항-c-fms 항체들에 대한 마커를 제공한다. Trap5b는 용골세포들에 의해 특이적으로 발현되며, 용골세포 수의 표시자(indicator)이다. 조혈세포(hematopoietic cells)들의 골수성 계열(myeloid lineage)로부터 유도되는 용골세포들은 c-fms를 발현하고 그리고 CSF-1을 활용한다. CSF-1 넉아웃(op/op) 생쥐들에서의 감소된 용골세포들 및 Trap5b의 수준들에서의 CSF-1 손실의 결과들이 이와 일치한다(다이 엑스엠과 그의 동료들의 Blood (2002) 99: 111 -120).
본트랩®(BoneTRAP®) 분석(미합중국 아리조나주 파운틴힐즈 소재의 임뮤노다이애그노스틱 시스템즈 리미티드(Immunodi agnostic Systems Limited))를 사용하여 대상체들에서의 TRAP5b를 정량하였다. 항체 1.2 SM 혈청 농도들을 앞서 기술한 바에 따라 결정하였다. 혈청 내에서의 TRAP5b 및 항체 1.2 SM의 수준들을 -7, 8, 29, 57, 85 및 99일 들에서 수집한 혈청 시료들에 대하여 결정하였다.
투여단계(dosing phase)의 29일차에서, 1.2 SM 항체로 처치된 모든 대상체들은 감소된 Trap5b를 가졌다. 후속하는 항체 1.2 SM으로의 처치로, 혈청 Trap5b 농도들은 혈청 항체 농도들이 감소함에 따라 증가하였다. 항체 1.2 SM으로의 처치는 감소된 혈청 Trap5b 농도와 연관되었다.
항체 1.2 SM으로의 처치에 대한 마크로파지의 응답
사이노몰거스 원숭이들에서의 항체 1.2 SM의 활성의 부가의 표시자로서, 결장 조직 내에 존재하는 마크로파지들의 수를 CD-68-염색된 조직의 레이저 주사 혈구계산(laser scanning cytometry ; LSC)에 의하여 정량하였다. 29일차 부검에서의 3개체의 동물들/성별/군(3 animals/sex/group) 및 100일차 부검에서의 2개체의 동물들/성별/군들로부터 결장 샘플들을 수집하였다. 각 조직의 샘플을 OCT(적정 절단 온도 ; Optimal Cutting Temperature) 배지(미합중국 캘리포니아주 토런스 소재의 사쿠라 파인테크(Sakura Finetek)) 내에서 수집하였으며, 드라이아이스/부탄 욕조(dry ice/butane bath) 내에서 동결시켰다. 항-CD68 또는 아이소타입 항체에 대하여 통상적인 면역조직화학법을 사용하여 마크로파지들을 염색시켰다. 디아미노벤지딘 양성 사건(diaminobenzidine(DAB) positive events)들을 레이저 주사 혈구계산(LSC)을 사용하여 열거하였다. 레이저 광 흡수가 스테이지(stage) 상의 광다이오드 검출기(photodiode detector)에 의해 정량화되는 2주사법(2 scan method)가 사용되었다. 첫 번째의 저해상도 통과가 슬라이드 상의 영역의 위치를 확인하고 그리고 후속하는 고해상도 통과가 필드이미지(field images)들을 획득하였다. 상기 LSC-연관 아이사이트 소프트웨어(iCyte software)를 사용하여 DAB 염색의 정량적인 분석을 수행하였다.
표 25(마크로파지 개체수에 대한 항체 1.2 SM의 영향)는 처치 직후(29일차) 및 항체 1.2 SM이 더 이상 투여되지 않는 후속하는 회복기간(99일차)의 결장 마크로파지의 수에서의 변화를 요약하고 있다. 이 표로부터 알 수 있는 바와 같이, 항체 1.2 SM의 투여는 콜로니 마크로파지(colony macrophage)의 수를 감소시킨 반면에, 처치가 연속되지 않은 후에는 마크로파지의 수가 증가하였으며, 따라서 상기 항체의 활성을 입증하고 있다.
| 분산분석(ANOVA) | 비교 그룹 | |||||
| 일수 | 효과 | p-값 | 투여량 (㎎/㎏) |
그룹 평균 | %감소 (대조 대비) |
p-값 (대조 대비) |
| 29 | 성별 | 0.4461 | 0 | 11.05 | ||
| 투여량 | 0.0005 | 20 | 1.75 | 84.16% | 0.0013 | |
| 100 | 2.36 | 78.64% | 0.0037 | |||
| 300 | 1.94 | 82.44% | 0.0003 | |||
| 99 | 성별 | 0.8172 | 0 | 10.62 | ||
| 투여량 | 0.1043 | 20 | 12.12 | -14.12% | 0.9953 | |
| 100 | 9.23 | 13.09% | 0.8493 | |||
| 300 | 2.67 | 74.86% | 0.0975 | |||
실시예
19 : c-
fms
결합 단백질을 사용하는
환자에서의
종양의 치료
인간 환자가 악성 종양을 앓는 것으로 진단되었다. 상기 환자를 본 명세서에서 기술된 c-fms 결합 단백질의 유효량으로 치료하였다. 상기 c-fms 결합 단백질의 투여 후, 상기 종양의 크기 및/또는 대사 활성을 측정하였다(예를 들면, MRI 또는 PET 주사들에 의하여). 상기 c-fms 결합 단백질의 투여에 대응하여 크기 및/또는 대사 활성 또는 종양 성장, 생존 능력, 전이 등의 다른 표시자들에서의 명백한 감소가 발견되었다.
실시예 20 : c-fms 결합 단백질을 사용하는 악성 종양을 앓는 환자에서의 TAMs의 감소
인간 환자가 악성 종양을 앓고 있는 것으로 진단되었다. 상기 환자를 본 명세서에서 기술된 c-fms 결합 단백질의 유효량으로 치료하였다. 상기 c-fms 결합 단백질의 투여 후, TAMs의 수를 측정하였다. 상기 c-fms 결합 단백질의 투여에 대응하여 TAMs의 수에서의 명백한 감소가 발견되었다.
실시예 21 : c-fms 결합 단백질을 사용하는 악액질의 치료
인간 환자가 암을 앓고 있는 것으로 진단되었다. 상기 환자를 본 명세서에서 기술된 c-fms 결합 단백질의 유효량으로 치료하였다. 상기 c-fms 결합 단백질의 투여 후, 악액질의 수준을 평가하였다. 상기 c-fms 결합 단백질의 투여에 대응하여 악액질의 수준에서의 명백한 감소가 발견되었다.
실시예 22 : c-fms 결합 단백질을 사용하는 혈관신생(vascularization)의 감소
인간 환자가 악성 종양을 앓고 있는 것으로 진단되었다. 상기 환자를 본 명세서에서 기술된 c-fms 결합 단백질의 유효량으로 치료하였다. 상기 c-fms 결합 단백질의 투여 후, 상기 종양의 생체검사를 하였으며, 혈관신생의 수준을 평가하였다. 상기 c-fms 결합 단백질의 투여에 대응하여 종양 혈관신생의 수준 및/또는 기능에서의 명백한 감소가 발견되었다.
실시예 23 : c-fms 결합 단백질을 사용하는 염증성 관절염의 치료
인간 환자가 염증성 관절염을 앓고 있는 것으로 진단되었다. 상기 환자를 본 명세서에서 기술된 c-fms 결합 단백질의 유효량으로 치료하였다. 상기 c-fms 결합 단백질의 투여 후, 염증의 수준 및/또는 골 밀도를 평가하였다. 상기 c-fms 결합 단백질의 투여에 대응하여 염증의 수준 및/또는 골 용해에서의 명백한 감소가 발견되었다.
확인된 모든 특허들 및 다른 간행물들은 기술되고 그리고 개시되는 목적, 예를 들면, 기술된 것들과 함께 사용될 수 있는 이러한 간행물들에서 기술된 방법론들에 대하여 본 명세서에서 참조로 인용되었다. 이들 간행물들은 본 출원의 출원일에 앞서 그들의 상세에 대하여 전적으로 제공되었다. 이와 관련하여 선행 발명의 덕분으로 또는 임의의 다른 이유로 선행하는 것으로 본 발명자들이 인정하는 것으로 이해되어야 할 것은 아무 것도 없다. 이들 문헌들의 날짜에 대한 것으로서의 언급 또는 내용들에 대한 것으로서의 묘사의 모든 것은 출원인들에게 획득가능한 정보에 기반한 것이며, 이들 문헌들의 날짜들 또는 내용들의 정확성에 대하여 어떠한 인정을 구성하는 것은 아니다.
<110> Amgen, Inc.
Kenneth Allan Brasel
James F. Smothers
Douglas Pat Cerretti
Christopher Mehlin
Jilin Sun
Stephen Foster
<120> HUMAN C-FMS ANTIGEN BINDING PROTEINS
<130> APMOL.007VPC
<150> 60/957,148
<151> 2007-08-21
<150> 61/084,588
<151> 2008-07-29
<160> 326
<170> FastSEQ for Windows Version 4.0
<210> 1
<211> 512
<212> PRT
<213> Homo Sapiens
<400> 1
Met Gly Pro Gly Val Leu Leu Leu Leu Leu Val Ala Thr Ala Trp His
1 5 10 15
Gly Gln Gly Ile Pro Val Ile Glu Pro Ser Val Pro Glu Leu Val Val
20 25 30
Lys Pro Gly Ala Thr Val Thr Leu Arg Cys Val Gly Asn Gly Ser Val
35 40 45
Glu Trp Asp Gly Pro Pro Ser Pro His Trp Thr Leu Tyr Ser Asp Gly
50 55 60
Ser Ser Ser Ile Leu Ser Thr Asn Asn Ala Thr Phe Gln Asn Thr Gly
65 70 75 80
Thr Tyr Arg Cys Thr Glu Pro Gly Asp Pro Leu Gly Gly Ser Ala Ala
85 90 95
Ile His Leu Tyr Val Lys Asp Pro Ala Arg Pro Trp Asn Val Leu Ala
100 105 110
Gln Glu Val Val Val Phe Glu Asp Gln Asp Ala Leu Leu Pro Cys Leu
115 120 125
Leu Thr Asp Pro Val Leu Glu Ala Gly Val Ser Leu Val Arg Val Arg
130 135 140
Gly Arg Pro Leu Met Arg His Thr Asn Tyr Ser Phe Ser Pro Trp His
145 150 155 160
Gly Phe Thr Ile His Arg Ala Lys Phe Ile Gln Ser Gln Asp Tyr Gln
165 170 175
Cys Ser Ala Leu Met Gly Gly Arg Lys Val Met Ser Ile Ser Ile Arg
180 185 190
Leu Lys Val Gln Lys Val Ile Pro Gly Pro Pro Ala Leu Thr Leu Val
195 200 205
Pro Ala Glu Leu Val Arg Ile Arg Gly Glu Ala Ala Gln Ile Val Cys
210 215 220
Ser Ala Ser Ser Val Asp Val Asn Phe Asp Val Phe Leu Gln His Asn
225 230 235 240
Asn Thr Lys Leu Ala Ile Pro Gln Gln Ser Asp Phe His Asn Asn Arg
245 250 255
Tyr Gln Lys Val Leu Thr Leu Asn Leu Asp Gln Val Asp Phe Gln His
260 265 270
Ala Gly Asn Tyr Ser Cys Val Ala Ser Asn Val Gln Gly Lys His Ser
275 280 285
Thr Ser Met Phe Phe Arg Val Val Glu Ser Ala Tyr Leu Asn Leu Ser
290 295 300
Ser Glu Gln Asn Leu Ile Gln Glu Val Thr Val Gly Glu Gly Leu Asn
305 310 315 320
Leu Lys Val Met Val Glu Ala Tyr Pro Gly Leu Gln Gly Phe Asn Trp
325 330 335
Thr Tyr Leu Gly Pro Phe Ser Asp His Gln Pro Glu Pro Lys Leu Ala
340 345 350
Asn Ala Thr Thr Lys Asp Thr Tyr Arg His Thr Phe Thr Leu Ser Leu
355 360 365
Pro Arg Leu Lys Pro Ser Glu Ala Gly Arg Tyr Ser Phe Leu Ala Arg
370 375 380
Asn Pro Gly Gly Trp Arg Ala Leu Thr Phe Glu Leu Thr Leu Arg Tyr
385 390 395 400
Pro Pro Glu Val Ser Val Ile Trp Thr Phe Ile Asn Gly Ser Gly Thr
405 410 415
Leu Leu Cys Ala Ala Ser Gly Tyr Pro Gln Pro Asn Val Thr Trp Leu
420 425 430
Gln Cys Ser Gly His Thr Asp Arg Cys Asp Glu Ala Gln Val Leu Gln
435 440 445
Val Trp Asp Asp Pro Tyr Pro Glu Val Leu Ser Gln Glu Pro Phe His
450 455 460
Lys Val Thr Val Gln Ser Leu Leu Thr Val Glu Thr Leu Glu His Asn
465 470 475 480
Gln Thr Tyr Glu Cys Arg Ala His Asn Ser Val Gly Ser Gly Ser Trp
485 490 495
Ala Phe Ile Pro Ile Ser Ala Gly Ala His Thr His Pro Pro Asp Glu
500 505 510
<210> 2
<211> 326
<212> PRT
<213> Homo Sapiens
<400> 2
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln Thr
65 70 75 80
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Thr Val Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro
100 105 110
Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
115 120 125
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
130 135 140
Val Ser His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly
145 150 155 160
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn
165 170 175
Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Val His Gln Asp Trp
180 185 190
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro
195 200 205
Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu
210 215 220
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
225 230 235 240
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
245 250 255
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
260 265 270
Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
275 280 285
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
290 295 300
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
305 310 315 320
Ser Leu Ser Pro Gly Lys
325
<210> 3
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 3
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
1 5 10 15
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
20 25 30
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
35 40 45
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
50 55 60
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
65 70 75 80
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
85 90 95
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
100 105
<210> 4
<211> 451
<212> PRT
<213> Homo Sapiens
<400> 4
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ala Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Pro Asn Ser Gly Gly Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Tyr Ser Gly Tyr Asp Leu Gly Tyr Tyr Tyr Gly Met
100 105 110
Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr
115 120 125
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser
130 135 140
Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
145 150 155 160
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
165 170 175
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
180 185 190
Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys
195 200 205
Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys Thr Val Glu
210 215 220
Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro Pro Val Ala
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe
290 295 300
Arg Val Val Ser Val Leu Thr Val Val His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 5
<211> 447
<212> PRT
<213> Homo Sapiens
<400> 5
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Val Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Asn Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Ala Gly Ile Ala Ala Thr Gly Thr Leu Phe Asp Cys Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn Val Asp His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg Lys Cys Cys
210 215 220
Val Glu Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val
225 230 235 240
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
245 250 255
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
260 265 270
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
275 280 285
Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg Val Val Ser
290 295 300
Val Leu Thr Val Val His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
305 310 315 320
Cys Lys Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu Lys Thr Ile
325 330 335
Ser Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
340 345 350
Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
355 360 365
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
370 375 380
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Met Leu Asp Ser
385 390 395 400
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
405 410 415
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
420 425 430
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 6
<211> 452
<212> PRT
<213> Homo Sapiens
<400> 6
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Pro Asn Ser Gly Gly Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Gly Gln Leu Trp Leu Trp Tyr Tyr Tyr Tyr Tyr Gly
100 105 110
Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser
115 120 125
Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr
130 135 140
Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro
145 150 155 160
Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val
165 170 175
His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser
180 185 190
Ser Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln Thr Tyr Thr
195 200 205
Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys Thr Val
210 215 220
Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro Pro Val
225 230 235 240
Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
245 250 255
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
260 265 270
His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu
275 280 285
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr
290 295 300
Phe Arg Val Val Ser Val Leu Thr Val Val His Gln Asp Trp Leu Asn
305 310 315 320
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ala Pro
325 330 335
Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln
340 345 350
Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val
355 360 365
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
370 375 380
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
385 390 395 400
Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
405 410 415
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
420 425 430
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
435 440 445
Ser Pro Gly Lys
450
<210> 7
<211> 445
<212> PRT
<213> Homo Sapiens
<400> 7
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ser Ser Ser Trp Ser Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly
100 105 110
Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn Val Asp His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg Lys Cys Cys Val Glu
210 215 220
Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Gln
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg Val Val Ser Val Leu
290 295 300
Thr Val Val His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys
325 330 335
Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Met Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 8
<211> 455
<212> PRT
<213> Homo Sapiens
<400> 8
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Val Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Asn Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Gly Gly Ser Leu Leu Trp Thr Gly Pro Asn Tyr Tyr
100 105 110
Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser
130 135 140
Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
145 150 155 160
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
165 170 175
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
180 185 190
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln
195 200 205
Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp
210 215 220
Lys Thr Val Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala
225 230 235 240
Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
245 250 255
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
260 265 270
Asp Val Ser His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
275 280 285
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
290 295 300
Asn Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Val His Gln Asp
305 310 315 320
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
325 330 335
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg
340 345 350
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys
355 360 365
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
370 375 380
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
385 390 395 400
Thr Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
405 410 415
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
420 425 430
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
435 440 445
Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 9
<211> 452
<212> PRT
<213> Homo Sapiens
<400> 9
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Glu Tyr Tyr Gly Ser Gly Gly Val Trp Tyr Tyr Gly
100 105 110
Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser
115 120 125
Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr
130 135 140
Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro
145 150 155 160
Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val
165 170 175
His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser
180 185 190
Ser Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln Thr Tyr Thr
195 200 205
Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys Thr Val
210 215 220
Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro Pro Val
225 230 235 240
Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
245 250 255
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
260 265 270
His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu
275 280 285
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr
290 295 300
Phe Arg Val Val Ser Val Leu Thr Val Val His Gln Asp Trp Leu Asn
305 310 315 320
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ala Pro
325 330 335
Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln
340 345 350
Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val
355 360 365
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
370 375 380
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
385 390 395 400
Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
405 410 415
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
420 425 430
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
435 440 445
Ser Pro Gly Lys
450
<210> 10
<211> 455
<212> PRT
<213> Homo Sapiens
<400> 10
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Trp Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Asp Leu Arg Ile Thr Gly Thr Thr Tyr Tyr Tyr Tyr
100 105 110
Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser
130 135 140
Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
145 150 155 160
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
165 170 175
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
180 185 190
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln
195 200 205
Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp
210 215 220
Lys Thr Val Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala
225 230 235 240
Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
245 250 255
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
260 265 270
Asp Val Ser His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
275 280 285
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
290 295 300
Asn Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Val His Gln Asp
305 310 315 320
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
325 330 335
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg
340 345 350
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys
355 360 365
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
370 375 380
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
385 390 395 400
Thr Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
405 410 415
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
420 425 430
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
435 440 445
Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 11
<211> 446
<212> PRT
<213> Homo Sapiens
<400> 11
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Ser Trp Phe Gly Glu Val Phe Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn Val Asp His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg Lys Cys Cys Val
210 215 220
Glu Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
260 265 270
Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg Val Val Ser Val
290 295 300
Leu Thr Val Val His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
325 330 335
Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Met Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 12
<211> 452
<212> PRT
<213> Homo Sapiens
<400> 12
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Glu Tyr Tyr Gly Ser Gly Gly Val Trp Tyr Tyr Gly
100 105 110
Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser
115 120 125
Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr
130 135 140
Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro
145 150 155 160
Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val
165 170 175
His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser
180 185 190
Ser Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln Thr Tyr Thr
195 200 205
Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys Thr Val
210 215 220
Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro Pro Val
225 230 235 240
Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
245 250 255
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
260 265 270
His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu
275 280 285
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr
290 295 300
Phe Arg Val Val Ser Val Leu Thr Val Val His Gln Asp Trp Leu Asn
305 310 315 320
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ala Pro
325 330 335
Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln
340 345 350
Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val
355 360 365
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
370 375 380
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
385 390 395 400
Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
405 410 415
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
420 425 430
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
435 440 445
Ser Pro Gly Lys
450
<210> 13
<211> 454
<212> PRT
<213> Homo Sapiens
<400> 13
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Asp Gly Ala Thr Val Val Thr Pro Gly Tyr Tyr Tyr
100 105 110
Tyr Gly Thr Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
130 135 140
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
145 150 155 160
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
165 170 175
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
180 185 190
Leu Ser Ser Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln Thr
195 200 205
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
210 215 220
Thr Val Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro
225 230 235 240
Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Ser His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn
290 295 300
Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Val His Gln Asp Trp
305 310 315 320
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro
325 330 335
Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu
340 345 350
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
355 360 365
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
370 375 380
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
385 390 395 400
Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
405 410 415
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
420 425 430
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
435 440 445
Ser Leu Ser Pro Gly Lys
450
<210> 14
<211> 446
<212> PRT
<213> Homo Sapiens
<400> 14
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Ser Trp Phe Gly Glu Val Phe Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn Val Asp His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg Lys Cys Cys Val
210 215 220
Glu Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
260 265 270
Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg Val Val Ser Val
290 295 300
Leu Thr Val Val His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
325 330 335
Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Met Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 15
<211> 446
<212> PRT
<213> Homo Sapiens
<400> 15
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Pro Asn Ser Gly Gly Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Phe Tyr Tyr Cys
85 90 95
Ala Arg Asp Ser Asn Trp Tyr His Asn Trp Phe Asp Pro Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn Val Asp His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg Lys Cys Cys Val
210 215 220
Glu Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
260 265 270
Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg Val Val Ser Val
290 295 300
Leu Thr Val Val His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
325 330 335
Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Met Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 16
<211> 446
<212> PRT
<213> Homo Sapiens
<400> 16
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Pro Asn Ser Gly Gly Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Phe Tyr Tyr Cys
85 90 95
Ala Arg Asp Ser Asn Trp Tyr His Asn Trp Phe Asp Pro Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn Val Asp His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg Lys Cys Cys Val
210 215 220
Glu Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
260 265 270
Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg Val Val Ser Val
290 295 300
Leu Thr Val Val His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
325 330 335
Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Met Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 17
<211> 454
<212> PRT
<213> Homo Sapiens
<400> 17
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Asp Gly Ala Thr Val Val Thr Pro Gly Tyr Tyr Tyr
100 105 110
Tyr Gly Thr Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
130 135 140
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
145 150 155 160
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
165 170 175
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
180 185 190
Leu Ser Ser Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln Thr
195 200 205
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
210 215 220
Thr Val Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro
225 230 235 240
Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Ser His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn
290 295 300
Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Val His Gln Asp Trp
305 310 315 320
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro
325 330 335
Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu
340 345 350
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
355 360 365
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
370 375 380
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
385 390 395 400
Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
405 410 415
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
420 425 430
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
435 440 445
Ser Leu Ser Pro Gly Lys
450
<210> 18
<211> 453
<212> PRT
<213> Homo Sapiens
<400> 18
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Ala Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Glu Gly Pro Tyr Ser Asp Tyr Gly Tyr Tyr Tyr Tyr
100 105 110
Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala
115 120 125
Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg Ser
130 135 140
Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe
145 150 155 160
Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly
165 170 175
Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu
180 185 190
Ser Ser Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln Thr Tyr
195 200 205
Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys Thr
210 215 220
Val Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro Pro
225 230 235 240
Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
245 250 255
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
260 265 270
Ser His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val
275 280 285
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser
290 295 300
Thr Phe Arg Val Val Ser Val Leu Thr Val Val His Gln Asp Trp Leu
305 310 315 320
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ala
325 330 335
Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu Pro
340 345 350
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
355 360 365
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
370 375 380
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
385 390 395 400
Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
405 410 415
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
420 425 430
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
435 440 445
Leu Ser Pro Gly Lys
450
<210> 19
<211> 446
<212> PRT
<213> Homo Sapiens
<400> 19
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Ser Trp Phe Gly Glu Val Phe Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn Val Asp His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg Lys Cys Cys Val
210 215 220
Glu Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
260 265 270
Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg Val Val Ser Val
290 295 300
Leu Thr Val Val His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
325 330 335
Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Met Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 20
<211> 443
<212> PRT
<213> Homo Sapiens
<400> 20
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Ala Thr Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Pro Gly Ser Val Lys
50 55 60
Gly Arg Phe Asn Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Gly Ser Trp Tyr Gly Phe Asp Tyr Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
115 120 125
Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys
130 135 140
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
145 150 155 160
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
165 170 175
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Asn
180 185 190
Phe Gly Thr Gln Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn
195 200 205
Thr Lys Val Asp Lys Thr Val Glu Arg Lys Cys Cys Val Glu Cys Pro
210 215 220
Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro
225 230 235 240
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
245 250 255
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Gln Phe Asn
260 265 270
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
275 280 285
Glu Glu Gln Phe Asn Ser Thr Phe Arg Val Val Ser Val Leu Thr Val
290 295 300
Val His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
305 310 315 320
Asn Lys Gly Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys
325 330 335
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu
340 345 350
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
355 360 365
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
370 375 380
Asn Asn Tyr Lys Thr Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe
385 390 395 400
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
405 410 415
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
420 425 430
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440
<210> 21
<211> 445
<212> PRT
<213> Homo Sapiens
<400> 21
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Asn Glu Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Ser Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala His Ser Ser Gly Asn Tyr Tyr Asp Met Asp Val Trp Gly Gln Gly
100 105 110
Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn Val Asp His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg Lys Cys Cys Val Glu
210 215 220
Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Gln
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg Val Val Ser Val Leu
290 295 300
Thr Val Val His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys
325 330 335
Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Met Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 22
<211> 453
<212> PRT
<213> Homo Sapiens
<400> 22
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Glu Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Asn Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Glu Gly Pro Tyr Ser Asn Tyr Gly Tyr Tyr Tyr Tyr
100 105 110
Gly Val Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala
115 120 125
Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg Ser
130 135 140
Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe
145 150 155 160
Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly
165 170 175
Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu
180 185 190
Ser Ser Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln Thr Tyr
195 200 205
Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys Thr
210 215 220
Val Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro Pro
225 230 235 240
Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
245 250 255
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
260 265 270
Ser His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val
275 280 285
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser
290 295 300
Thr Phe Arg Val Val Ser Val Leu Thr Val Val His Gln Asp Trp Leu
305 310 315 320
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ala
325 330 335
Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu Pro
340 345 350
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
355 360 365
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
370 375 380
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
385 390 395 400
Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
405 410 415
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
420 425 430
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
435 440 445
Leu Ser Pro Gly Lys
450
<210> 23
<211> 456
<212> PRT
<213> Homo Sapiens
<400> 23
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Thr Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Glu Tyr Tyr His Ile Leu Thr Gly Ser Phe Tyr Tyr
100 105 110
Ser Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val
115 120 125
Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys
130 135 140
Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys
145 150 155 160
Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu
165 170 175
Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu
180 185 190
Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr
195 200 205
Gln Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val
210 215 220
Asp Lys Thr Val Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro
225 230 235 240
Ala Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
245 250 255
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
260 265 270
Val Asp Val Ser His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val
275 280 285
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
290 295 300
Phe Asn Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Val His Gln
305 310 315 320
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly
325 330 335
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro
340 345 350
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr
355 360 365
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
370 375 380
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
385 390 395 400
Lys Thr Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
405 410 415
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
420 425 430
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
435 440 445
Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 24
<211> 445
<212> PRT
<213> Homo Sapiens
<400> 24
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ser Ser Ser Ser Asn Phe Tyr Asp Met Asp Val Trp Gly Gln Gly
100 105 110
Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn Val Asp His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg Lys Cys Cys Val Glu
210 215 220
Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Gln
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg Val Val Ser Val Leu
290 295 300
Thr Val Val His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys
325 330 335
Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Met Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 25
<211> 443
<212> PRT
<213> Homo Sapiens
<400> 25
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Asn Tyr
20 25 30
Tyr Trp Ser Trp Ile Arg Gln Ser Ala Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Arg Ile Tyr Thr Ser Gly Ser Thr His Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Ile Ile Met Ser Val Asp Thr Ser Lys Asn Gln Phe Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Asp Arg Val Phe Tyr Gly Met Asp Val Trp Gly Gln Gly Thr Thr
100 105 110
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
115 120 125
Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys
130 135 140
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
145 150 155 160
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
165 170 175
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Asn
180 185 190
Phe Gly Thr Gln Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn
195 200 205
Thr Lys Val Asp Lys Thr Val Glu Arg Lys Cys Cys Val Glu Cys Pro
210 215 220
Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro
225 230 235 240
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
245 250 255
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Gln Phe Asn
260 265 270
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
275 280 285
Glu Glu Gln Phe Asn Ser Thr Phe Arg Val Val Ser Val Leu Thr Val
290 295 300
Val His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
305 310 315 320
Asn Lys Gly Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys
325 330 335
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu
340 345 350
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
355 360 365
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
370 375 380
Asn Asn Tyr Lys Thr Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe
385 390 395 400
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
405 410 415
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
420 425 430
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440
<210> 26
<211> 447
<212> PRT
<213> Homo Sapiens
<400> 26
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Tyr Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Asp Tyr Ser Asp Tyr Tyr Gly Met Asp Val Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn Val Asp His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg Lys Cys Cys
210 215 220
Val Glu Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val
225 230 235 240
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
245 250 255
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
260 265 270
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
275 280 285
Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg Val Val Ser
290 295 300
Val Leu Thr Val Val His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
305 310 315 320
Cys Lys Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu Lys Thr Ile
325 330 335
Ser Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
340 345 350
Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
355 360 365
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
370 375 380
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Met Leu Asp Ser
385 390 395 400
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
405 410 415
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
420 425 430
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 27
<211> 455
<212> PRT
<213> Homo Sapiens
<400> 27
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Gly Val Met Ile Thr Phe Gly Gly Val Ile Val Gly His Ser
100 105 110
Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser
130 135 140
Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
145 150 155 160
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
165 170 175
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
180 185 190
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln
195 200 205
Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp
210 215 220
Lys Thr Val Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala
225 230 235 240
Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
245 250 255
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
260 265 270
Asp Val Ser His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
275 280 285
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
290 295 300
Asn Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Val His Gln Asp
305 310 315 320
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
325 330 335
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg
340 345 350
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys
355 360 365
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
370 375 380
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
385 390 395 400
Thr Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
405 410 415
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
420 425 430
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
435 440 445
Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 28
<211> 455
<212> PRT
<213> Homo Sapiens
<400> 28
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Gly Val Met Ile Thr Phe Gly Gly Val Ile Val Gly His Ser
100 105 110
Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser
130 135 140
Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
145 150 155 160
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
165 170 175
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
180 185 190
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln
195 200 205
Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp
210 215 220
Lys Thr Val Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala
225 230 235 240
Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
245 250 255
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
260 265 270
Asp Val Ser His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
275 280 285
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
290 295 300
Asn Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Val His Gln Asp
305 310 315 320
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
325 330 335
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg
340 345 350
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys
355 360 365
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
370 375 380
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
385 390 395 400
Thr Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
405 410 415
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
420 425 430
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
435 440 445
Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 29
<211> 450
<212> PRT
<213> Homo Sapiens
<400> 29
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Arg Ala Gly Thr Thr Leu Ala Tyr Tyr Tyr Tyr Ala Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys
115 120 125
Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu
130 135 140
Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
145 150 155 160
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
165 170 175
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
180 185 190
Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn
195 200 205
Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg
210 215 220
Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg
290 295 300
Val Val Ser Val Leu Thr Val Val His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Met
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 30
<211> 442
<212> PRT
<213> Homo Sapiens
<400> 30
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Tyr
20 25 30
Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Tyr Tyr Ser Gly Asn Thr Asn Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Phe Thr Leu Ser Ile Asp Thr Ser Lys Asn Gln Phe Ser Leu
65 70 75 80
Arg Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Cys Ile Ala Thr Arg Pro Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val
100 105 110
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
115 120 125
Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu
130 135 140
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
145 150 155 160
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
165 170 175
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Asn Phe
180 185 190
Gly Thr Gln Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr
195 200 205
Lys Val Asp Lys Thr Val Glu Arg Lys Cys Cys Val Glu Cys Pro Pro
210 215 220
Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro
225 230 235 240
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
245 250 255
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Gln Phe Asn Trp
260 265 270
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
275 280 285
Glu Gln Phe Asn Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Val
290 295 300
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
305 310 315 320
Lys Gly Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly
325 330 335
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu
340 345 350
Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
355 360 365
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
370 375 380
Asn Tyr Lys Thr Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe
385 390 395 400
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
405 410 415
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
420 425 430
Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440
<210> 31
<211> 445
<212> PRT
<213> Homo Sapiens
<400> 31
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ile Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Asp Ser Ser Gly Asp Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly
100 105 110
Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn Val Asp His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg Lys Cys Cys Val Glu
210 215 220
Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Gln
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg Val Val Ser Val Leu
290 295 300
Thr Val Val His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys
325 330 335
Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Met Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 32
<211> 445
<212> PRT
<213> Homo Sapiens
<400> 32
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Ala Gly Leu Glu Ile Arg Trp Phe Asp Pro Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn Val Asp His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg Lys Cys Cys Val Glu
210 215 220
Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Gln
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg Val Val Ser Val Leu
290 295 300
Thr Val Val His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys
325 330 335
Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Met Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 33
<211> 446
<212> PRT
<213> Homo Sapiens
<400> 33
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln His Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Ser Tyr Ser Gly Asp Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Leu Thr Ile Ser Val Asp Thr Ser Lys His Gln Phe
65 70 75 80
Ser Leu Arg Leu Ser Ser Val Thr Ser Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Ser Leu Asp Leu Tyr Gly Asp Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn Val Asp His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg Lys Cys Cys Val
210 215 220
Glu Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
260 265 270
Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg Val Val Ser Val
290 295 300
Leu Thr Val Val His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
325 330 335
Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Met Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 34
<211> 451
<212> PRT
<213> Homo Sapiens
<400> 34
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Leu Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Pro Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Gln Gly Leu Leu Gly Phe Gly Glu Leu Glu Gly Leu Phe
100 105 110
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr
115 120 125
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser
130 135 140
Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
145 150 155 160
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
165 170 175
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
180 185 190
Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys
195 200 205
Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys Thr Val Glu
210 215 220
Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro Pro Val Ala
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe
290 295 300
Arg Val Val Ser Val Leu Thr Val Val His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 35
<211> 456
<212> PRT
<213> Homo Sapiens
<400> 35
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Thr Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Gln Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Glu Tyr Tyr Gly Ile Val Thr Gly Ser Phe Tyr Tyr
100 105 110
Tyr Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val
115 120 125
Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys
130 135 140
Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys
145 150 155 160
Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu
165 170 175
Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu
180 185 190
Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr
195 200 205
Gln Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val
210 215 220
Asp Lys Thr Val Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro
225 230 235 240
Ala Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
245 250 255
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
260 265 270
Val Asp Val Ser His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val
275 280 285
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
290 295 300
Phe Asn Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Val His Gln
305 310 315 320
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly
325 330 335
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro
340 345 350
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr
355 360 365
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
370 375 380
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
385 390 395 400
Lys Thr Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
405 410 415
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
420 425 430
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
435 440 445
Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 36
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 36
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asn Ile Ser Asn Phe
20 25 30
Leu Asp Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Asn Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asp Leu Asp Pro Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Val Ser Leu Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 37
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 37
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Phe
20 25 30
Leu Asp Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asp Leu Asp Pro Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Val Ser Leu Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 38
<211> 219
<212> PRT
<213> Homo Sapiens
<400> 38
Asp Asn Val Met Thr Gln Thr Pro Leu Ser Leu Ser Val Thr Pro Gly
1 5 10 15
Gln Pro Ala Ser Ile Ser Cys Lys Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asp Gly Lys Thr Tyr Leu Tyr Trp Tyr Leu Gln Lys Pro Gly Gln Pro
35 40 45
Pro Gln Leu Leu Ile Tyr Glu Ala Ser Asn Arg Phe Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ser
85 90 95
Ile Gln Leu Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 39
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 39
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Asn Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Ile Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Ile Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Phe Pro Phe
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 40
<211> 213
<212> PRT
<213> Homo Sapiens
<400> 40
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Asn Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Thr Ser Asn Leu Glu Pro Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Leu Thr
85 90 95
Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg Thr Val Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys
210
<210> 41
<211> 213
<212> PRT
<213> Homo Sapiens
<400> 41
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln His Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Leu Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys
210
<210> 42
<211> 213
<212> PRT
<213> Homo Sapiens
<400> 42
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Phe Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Ile Thr
85 90 95
Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg Thr Val Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys
210
<210> 43
<211> 220
<212> PRT
<213> Homo Sapiens
<400> 43
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Val Leu Asp Ser
20 25 30
Ser Asp Asn Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Asn Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Ser Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Asp Pro Phe Thr Phe Gly Pro Gly Thr Lys Val Asp Ile
100 105 110
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
115 120 125
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
130 135 140
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
145 150 155 160
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
165 170 175
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
180 185 190
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
195 200 205
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 44
<211> 220
<212> PRT
<213> Homo Sapiens
<400> 44
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Val Leu Asp Ser
20 25 30
Ser Asp Asn Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Asn Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Asp Pro Phe Thr Phe Gly Pro Gly Thr Lys Val Asp Ile
100 105 110
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
115 120 125
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
130 135 140
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
145 150 155 160
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
165 170 175
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
180 185 190
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
195 200 205
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 45
<211> 213
<212> PRT
<213> Homo Sapiens
<400> 45
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Leu Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys
210
<210> 46
<211> 220
<212> PRT
<213> Homo Sapiens
<400> 46
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asp Cys Lys Ser Ser Gln Gly Val Leu Asp Ser
20 25 30
Ser Asn Asn Lys Asn Phe Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Asn Arg Glu Ser Gly Val
50 55 60
Pro Val Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Leu Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Asp Pro Phe Thr Phe Gly Pro Gly Thr Lys Val Asp Ile
100 105 110
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
115 120 125
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
130 135 140
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
145 150 155 160
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
165 170 175
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
180 185 190
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
195 200 205
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 47
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 47
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Asp Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Asn Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Phe Cys Gln Gln Thr Tyr Ser Asp Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 48
<211> 213
<212> PRT
<213> Homo Sapiens
<400> 48
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Leu Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys
210
<210> 49
<211> 213
<212> PRT
<213> Homo Sapiens
<400> 49
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Leu Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys
210
<210> 50
<211> 220
<212> PRT
<213> Homo Sapiens
<400> 50
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asp Cys Lys Ser Ser Gln Ser Val Leu Asp Ser
20 25 30
Ser Asn Asn Lys Asn Phe Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Asn Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Asp Pro Phe Thr Phe Gly Pro Gly Thr Lys Val Asp Ile
100 105 110
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
115 120 125
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
130 135 140
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
145 150 155 160
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
165 170 175
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
180 185 190
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
195 200 205
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 51
<211> 218
<212> PRT
<213> Homo Sapiens
<400> 51
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Gly
20 25 30
Tyr Leu Ala Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
35 40 45
Arg Leu Leu Ile Tyr Gly Ala Ser Ser Thr Ala Thr Gly Ile Pro Asp
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly
85 90 95
Ser Ser Pro Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 52
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 52
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Phe
20 25 30
Leu Asn Trp Tyr Gln Gln Arg Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 53
<211> 213
<212> PRT
<213> Homo Sapiens
<400> 53
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Leu Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys
210
<210> 54
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 54
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Arg Tyr Asp Asp Leu Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 55
<211> 213
<212> PRT
<213> Homo Sapiens
<400> 55
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Arg Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Leu Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys
210
<210> 56
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 56
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Phe Ser Asn Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Thr Tyr Tyr Cys Gln Lys Tyr Asn Ser Ala Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 57
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 57
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Asn Asn Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Gln Leu Leu Ile
35 40 45
Tyr Val Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Thr Tyr Tyr Cys Gln Lys Tyr Asn Ser Gly Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 58
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 58
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Asn Asn Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Thr Tyr Tyr Cys Gln Lys Tyr Asn Ser Gly Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 59
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 59
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Arg Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Asn Leu Leu Ile
35 40 45
His Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ile Thr Pro Pro
85 90 95
Ser Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 60
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 60
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Arg Asn Asp
20 25 30
Leu Asp Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Asn Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Tyr Asn Ser Tyr Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 61
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 61
Asp Ile Gln Met Ile Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser His Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Phe Leu Ile
35 40 45
Ser Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 62
<211> 219
<212> PRT
<213> Homo Sapiens
<400> 62
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Phe Leu Ile Tyr Leu Gly Ser Ile Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Ala Leu Thr Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Arg Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 63
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 63
Glu Ile Val Leu Thr Gln Ser Pro Asp Phe Gln Ser Val Thr Pro Lys
1 5 10 15
Glu Lys Val Thr Ile Thr Cys Arg Ala Ser Gln Tyr Ile Gly Ser Ser
20 25 30
Leu His Trp Tyr Gln Gln Thr Pro Asp Gln Ser Pro Lys Leu Leu Ile
35 40 45
Asn Tyr Val Ser Gln Ser Phe Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Asn Ser Leu Glu Ala
65 70 75 80
Glu Asp Ala Ala Thr Tyr Tyr Cys His Gln Ser Ser Ser Leu Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 64
<211> 220
<212> PRT
<213> Homo Sapiens
<400> 64
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Ala Arg Ala Thr Ile Ser Cys Lys Ser Ser Gln Ser Val Leu Tyr Ser
20 25 30
Ser Asn Asn Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Thr Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Thr Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
100 105 110
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
115 120 125
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
130 135 140
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
145 150 155 160
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
165 170 175
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
180 185 190
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
195 200 205
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 65
<211> 213
<212> PRT
<213> Homo Sapiens
<400> 65
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Asn Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asp Leu Leu Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys
210
<210> 66
<211> 214
<212> PRT
<213> Homo Sapiens
<400> 66
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Ile Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Phe Asp Asn Leu Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ser Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 67
<211> 213
<212> PRT
<213> Homo Sapiens
<400> 67
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln His Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Leu Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys
210
<210> 68
<211> 222
<212> PRT
<213> Homo Sapiens
<400> 68
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Leu Gly
1 5 10 15
Gln Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Val Tyr Ser
20 25 30
Asp Gly Asn Thr Tyr Leu Asn Trp Phe Gln Gln Arg Pro Gly Gln Ser
35 40 45
Pro Arg Arg Leu Ile Tyr Lys Val Ser Asn Trp Asp Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Gly
85 90 95
Thr His Trp Pro Arg Gly Leu Phe Thr Phe Gly Pro Gly Thr Lys Val
100 105 110
Asp Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
115 120 125
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
130 135 140
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
145 150 155 160
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
165 170 175
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
180 185 190
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
195 200 205
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 69
<211> 219
<212> PRT
<213> Homo Sapiens
<400> 69
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Leu Gly
1 5 10 15
Gln Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Val Tyr Ser
20 25 30
Asp Gly Asn Thr Tyr Leu Asn Trp Phe Gln Gln Arg Pro Gly Gln Ser
35 40 45
Pro Arg Arg Leu Ile Tyr Lys Val Ser Asn Trp Asp Ser Gly Val Pro
50 55 60
Asp Arg Phe Asn Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Gly
85 90 95
Thr His Trp Pro Ile Thr Phe Gly Gln Gly Thr Gly Leu Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 70
<211> 125
<212> PRT
<213> Homo Sapiens
<400> 70
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ala Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Pro Asn Ser Gly Gly Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Tyr Ser Gly Tyr Asp Leu Gly Tyr Tyr Tyr Gly Met
100 105 110
Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 71
<211> 121
<212> PRT
<213> Homo Sapiens
<400> 71
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Val Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Asn Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Ala Gly Ile Ala Ala Thr Gly Thr Leu Phe Asp Cys Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 72
<211> 126
<212> PRT
<213> Homo Sapiens
<400> 72
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Pro Asn Ser Gly Gly Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Gly Gln Leu Trp Leu Trp Tyr Tyr Tyr Tyr Tyr Gly
100 105 110
Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 73
<211> 119
<212> PRT
<213> Homo Sapiens
<400> 73
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ser Ser Ser Trp Ser Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly
100 105 110
Thr Thr Val Thr Val Ser Ser
115
<210> 74
<211> 129
<212> PRT
<213> Homo Sapiens
<400> 74
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Val Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Asn Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Gly Gly Ser Leu Leu Trp Thr Gly Pro Asn Tyr Tyr
100 105 110
Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser
<210> 75
<211> 126
<212> PRT
<213> Homo Sapiens
<400> 75
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Glu Tyr Tyr Gly Ser Gly Gly Val Trp Tyr Tyr Gly
100 105 110
Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 76
<211> 129
<212> PRT
<213> Homo Sapiens
<400> 76
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Trp Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Asp Leu Arg Ile Thr Gly Thr Thr Tyr Tyr Tyr Tyr
100 105 110
Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser
<210> 77
<211> 120
<212> PRT
<213> Homo Sapiens
<400> 77
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Ser Trp Phe Gly Glu Val Phe Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 78
<211> 126
<212> PRT
<213> Homo Sapiens
<400> 78
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Glu Tyr Tyr Gly Ser Gly Gly Val Trp Tyr Tyr Gly
100 105 110
Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 79
<211> 128
<212> PRT
<213> Homo Sapiens
<400> 79
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Asp Gly Ala Thr Val Val Thr Pro Gly Tyr Tyr Tyr
100 105 110
Tyr Gly Thr Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 80
<211> 120
<212> PRT
<213> Homo Sapiens
<400> 80
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Ser Trp Phe Gly Glu Val Phe Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 81
<211> 120
<212> PRT
<213> Homo Sapiens
<400> 81
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Pro Asn Ser Gly Gly Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Phe Tyr Tyr Cys
85 90 95
Ala Arg Asp Ser Asn Trp Tyr His Asn Trp Phe Asp Pro Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 82
<211> 120
<212> PRT
<213> Homo Sapiens
<400> 82
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Pro Asn Ser Gly Gly Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Phe Tyr Tyr Cys
85 90 95
Ala Arg Asp Ser Asn Trp Tyr His Asn Trp Phe Asp Pro Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 83
<211> 128
<212> PRT
<213> Homo Sapiens
<400> 83
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Asp Gly Ala Thr Val Val Thr Pro Gly Tyr Tyr Tyr
100 105 110
Tyr Gly Thr Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 84
<211> 127
<212> PRT
<213> Homo Sapiens
<400> 84
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Ala Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Glu Gly Pro Tyr Ser Asp Tyr Gly Tyr Tyr Tyr Tyr
100 105 110
Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 85
<211> 120
<212> PRT
<213> Homo Sapiens
<400> 85
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Ser Trp Phe Gly Glu Val Phe Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 86
<211> 117
<212> PRT
<213> Homo Sapiens
<400> 86
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Ala Thr Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Pro Gly Ser Val Lys
50 55 60
Gly Arg Phe Asn Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Gly Ser Trp Tyr Gly Phe Asp Tyr Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser
115
<210> 87
<211> 119
<212> PRT
<213> Homo Sapiens
<400> 87
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Asn Glu Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Ser Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala His Ser Ser Gly Asn Tyr Tyr Asp Met Asp Val Trp Gly Gln Gly
100 105 110
Thr Thr Val Thr Val Ser Ser
115
<210> 88
<211> 127
<212> PRT
<213> Homo Sapiens
<400> 88
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Glu Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Asn Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Glu Gly Pro Tyr Ser Asn Tyr Gly Tyr Tyr Tyr Tyr
100 105 110
Gly Val Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 89
<211> 130
<212> PRT
<213> Homo Sapiens
<400> 89
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Thr Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Glu Tyr Tyr His Ile Leu Thr Gly Ser Phe Tyr Tyr
100 105 110
Ser Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val
115 120 125
Ser Ser
130
<210> 90
<211> 119
<212> PRT
<213> Homo Sapiens
<400> 90
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ser Ser Ser Ser Asn Phe Tyr Asp Met Asp Val Trp Gly Gln Gly
100 105 110
Thr Thr Val Thr Val Ser Ser
115
<210> 91
<211> 117
<212> PRT
<213> Homo Sapiens
<400> 91
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Asn Tyr
20 25 30
Tyr Trp Ser Trp Ile Arg Gln Ser Ala Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Arg Ile Tyr Thr Ser Gly Ser Thr His Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Ile Ile Met Ser Val Asp Thr Ser Lys Asn Gln Phe Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Asp Arg Val Phe Tyr Gly Met Asp Val Trp Gly Gln Gly Thr Thr
100 105 110
Val Thr Val Ser Ser
115
<210> 92
<211> 121
<212> PRT
<213> Homo Sapiens
<400> 92
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Tyr Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Asp Tyr Ser Asp Tyr Tyr Gly Met Asp Val Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 93
<211> 129
<212> PRT
<213> Homo Sapiens
<400> 93
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Gly Val Met Ile Thr Phe Gly Gly Val Ile Val Gly His Ser
100 105 110
Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser
<210> 94
<211> 129
<212> PRT
<213> Homo Sapiens
<400> 94
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Gly Val Met Ile Thr Phe Gly Gly Val Ile Val Gly His Ser
100 105 110
Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser
<210> 95
<211> 124
<212> PRT
<213> Homo Sapiens
<400> 95
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Arg Ala Gly Thr Thr Leu Ala Tyr Tyr Tyr Tyr Ala Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 96
<211> 116
<212> PRT
<213> Homo Sapiens
<400> 96
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Tyr
20 25 30
Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Tyr Tyr Ser Gly Asn Thr Asn Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Phe Thr Leu Ser Ile Asp Thr Ser Lys Asn Gln Phe Ser Leu
65 70 75 80
Arg Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Cys Ile Ala Thr Arg Pro Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val
100 105 110
Thr Val Ser Ser
115
<210> 97
<211> 119
<212> PRT
<213> Homo Sapiens
<400> 97
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ile Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Asp Ser Ser Gly Asp Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly
100 105 110
Thr Thr Val Thr Val Ser Ser
115
<210> 98
<211> 119
<212> PRT
<213> Homo Sapiens
<400> 98
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Ala Gly Leu Glu Ile Arg Trp Phe Asp Pro Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 99
<211> 120
<212> PRT
<213> Homo Sapiens
<400> 99
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Gly Tyr Tyr Trp Ser Trp Ile Arg Gln His Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Ser Tyr Ser Gly Asp Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Leu Thr Ile Ser Val Asp Thr Ser Lys His Gln Phe
65 70 75 80
Ser Leu Arg Leu Ser Ser Val Thr Ser Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Ser Leu Asp Leu Tyr Gly Asp Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 100
<211> 125
<212> PRT
<213> Homo Sapiens
<400> 100
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Leu Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Pro Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Gln Gly Leu Leu Gly Phe Gly Glu Leu Glu Gly Leu Phe
100 105 110
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 101
<211> 130
<212> PRT
<213> Homo Sapiens
<400> 101
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Thr Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Gln Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Glu Tyr Tyr Gly Ile Val Thr Gly Ser Phe Tyr Tyr
100 105 110
Tyr Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val
115 120 125
Ser Ser
130
<210> 102
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 102
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asn Ile Ser Asn Phe
20 25 30
Leu Asp Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Asn Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asp Leu Asp Pro Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Val Ser Leu Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 103
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 103
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Phe
20 25 30
Leu Asp Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asp Leu Asp Pro Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Val Ser Leu Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 104
<211> 112
<212> PRT
<213> Homo Sapiens
<400> 104
Asp Asn Val Met Thr Gln Thr Pro Leu Ser Leu Ser Val Thr Pro Gly
1 5 10 15
Gln Pro Ala Ser Ile Ser Cys Lys Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asp Gly Lys Thr Tyr Leu Tyr Trp Tyr Leu Gln Lys Pro Gly Gln Pro
35 40 45
Pro Gln Leu Leu Ile Tyr Glu Ala Ser Asn Arg Phe Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ser
85 90 95
Ile Gln Leu Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 105
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 105
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Asn Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Ile Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Ile Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Phe Pro Phe
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 106
<211> 106
<212> PRT
<213> Homo Sapiens
<400> 106
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Asn Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Thr Ser Asn Leu Glu Pro Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Leu Thr
85 90 95
Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 107
<211> 106
<212> PRT
<213> Homo Sapiens
<400> 107
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln His Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Leu Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 108
<211> 106
<212> PRT
<213> Homo Sapiens
<400> 108
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Phe Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Ile Thr
85 90 95
Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 109
<211> 113
<212> PRT
<213> Homo Sapiens
<400> 109
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Val Leu Asp Ser
20 25 30
Ser Asp Asn Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Asn Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Ser Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Asp Pro Phe Thr Phe Gly Pro Gly Thr Lys Val Asp Ile
100 105 110
Lys
<210> 110
<211> 113
<212> PRT
<213> Homo Sapiens
<400> 110
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Val Leu Asp Ser
20 25 30
Ser Asp Asn Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Asn Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Asp Pro Phe Thr Phe Gly Pro Gly Thr Lys Val Asp Ile
100 105 110
Lys
<210> 111
<211> 106
<212> PRT
<213> Homo Sapiens
<400> 111
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Leu Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 112
<211> 113
<212> PRT
<213> Homo Sapiens
<400> 112
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asp Cys Lys Ser Ser Gln Gly Val Leu Asp Ser
20 25 30
Ser Asn Asn Lys Asn Phe Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Asn Arg Glu Ser Gly Val
50 55 60
Pro Val Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Leu Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Asp Pro Phe Thr Phe Gly Pro Gly Thr Lys Val Asp Ile
100 105 110
Lys
<210> 113
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 113
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Asp Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Asn Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Phe Cys Gln Gln Thr Tyr Ser Asp Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 114
<211> 106
<212> PRT
<213> Homo Sapiens
<400> 114
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Leu Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 115
<211> 106
<212> PRT
<213> Homo Sapiens
<400> 115
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Leu Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 116
<211> 113
<212> PRT
<213> Homo Sapiens
<400> 116
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asp Cys Lys Ser Ser Gln Ser Val Leu Asp Ser
20 25 30
Ser Asn Asn Lys Asn Phe Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Asn Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Asp Pro Phe Thr Phe Gly Pro Gly Thr Lys Val Asp Ile
100 105 110
Lys
<210> 117
<211> 111
<212> PRT
<213> Homo Sapiens
<400> 117
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Gly
20 25 30
Tyr Leu Ala Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
35 40 45
Arg Leu Leu Ile Tyr Gly Ala Ser Ser Thr Ala Thr Gly Ile Pro Asp
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly
85 90 95
Ser Ser Pro Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105 110
<210> 118
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 118
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Phe
20 25 30
Leu Asn Trp Tyr Gln Gln Arg Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 119
<211> 106
<212> PRT
<213> Homo Sapiens
<400> 119
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Leu Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 120
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 120
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Arg Tyr Asp Asp Leu Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 121
<211> 106
<212> PRT
<213> Homo Sapiens
<400> 121
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Arg Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Leu Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 122
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 122
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Phe Ser Asn Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Thr Tyr Tyr Cys Gln Lys Tyr Asn Ser Ala Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 123
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 123
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Asn Asn Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Gln Leu Leu Ile
35 40 45
Tyr Val Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Thr Tyr Tyr Cys Gln Lys Tyr Asn Ser Gly Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 124
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 124
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Asn Asn Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Thr Tyr Tyr Cys Gln Lys Tyr Asn Ser Gly Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 125
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 125
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Arg Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Asn Leu Leu Ile
35 40 45
His Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ile Thr Pro Pro
85 90 95
Ser Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 126
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 126
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Arg Asn Asp
20 25 30
Leu Asp Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Asn Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Tyr Asn Ser Tyr Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 127
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 127
Asp Ile Gln Met Ile Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser His Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Phe Leu Ile
35 40 45
Ser Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 128
<211> 112
<212> PRT
<213> Homo Sapiens
<400> 128
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Phe Leu Ile Tyr Leu Gly Ser Ile Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Ala Leu Thr Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Arg Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 129
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 129
Glu Ile Val Leu Thr Gln Ser Pro Asp Phe Gln Ser Val Thr Pro Lys
1 5 10 15
Glu Lys Val Thr Ile Thr Cys Arg Ala Ser Gln Tyr Ile Gly Ser Ser
20 25 30
Leu His Trp Tyr Gln Gln Thr Pro Asp Gln Ser Pro Lys Leu Leu Ile
35 40 45
Asn Tyr Val Ser Gln Ser Phe Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Asn Ser Leu Glu Ala
65 70 75 80
Glu Asp Ala Ala Thr Tyr Tyr Cys His Gln Ser Ser Ser Leu Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 130
<211> 113
<212> PRT
<213> Homo Sapiens
<400> 130
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Ala Arg Ala Thr Ile Ser Cys Lys Ser Ser Gln Ser Val Leu Tyr Ser
20 25 30
Ser Asn Asn Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Thr Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Thr Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
100 105 110
Lys
<210> 131
<211> 106
<212> PRT
<213> Homo Sapiens
<400> 131
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Asn Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asp Leu Leu Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 132
<211> 107
<212> PRT
<213> Homo Sapiens
<400> 132
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Ile Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Phe Asp Asn Leu Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ser Lys
100 105
<210> 133
<211> 106
<212> PRT
<213> Homo Sapiens
<400> 133
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln His Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Leu Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 134
<211> 115
<212> PRT
<213> Homo Sapiens
<400> 134
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Leu Gly
1 5 10 15
Gln Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Val Tyr Ser
20 25 30
Asp Gly Asn Thr Tyr Leu Asn Trp Phe Gln Gln Arg Pro Gly Gln Ser
35 40 45
Pro Arg Arg Leu Ile Tyr Lys Val Ser Asn Trp Asp Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Gly
85 90 95
Thr His Trp Pro Arg Gly Leu Phe Thr Phe Gly Pro Gly Thr Lys Val
100 105 110
Asp Ile Lys
115
<210> 135
<211> 112
<212> PRT
<213> Homo Sapiens
<400> 135
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Leu Gly
1 5 10 15
Gln Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Val Tyr Ser
20 25 30
Asp Gly Asn Thr Tyr Leu Asn Trp Phe Gln Gln Arg Pro Gly Gln Ser
35 40 45
Pro Arg Arg Leu Ile Tyr Lys Val Ser Asn Trp Asp Ser Gly Val Pro
50 55 60
Asp Arg Phe Asn Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Gly
85 90 95
Thr His Trp Pro Ile Thr Phe Gly Gln Gly Thr Gly Leu Glu Ile Lys
100 105 110
<210> 136
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 136
Ser Gly Gly Tyr Tyr Trp Ser
1 5
<210> 137
<211> 5
<212> PRT
<213> Homo Sapiens
<400> 137
Asn Ala Trp Met Ser
1 5
<210> 138
<211> 5
<212> PRT
<213> Homo Sapiens
<400> 138
Thr Ala Trp Met Ser
1 5
<210> 139
<211> 5
<212> PRT
<213> Homo Sapiens
<400> 139
Gly Tyr Tyr Met His
1 5
<210> 140
<211> 5
<212> PRT
<213> Homo Sapiens
<400> 140
Ala Tyr Tyr Met His
1 5
<210> 141
<211> 5
<212> PRT
<213> Homo Sapiens
<400> 141
Ser Tyr Asp Met His
1 5
<210> 142
<211> 5
<212> PRT
<213> Homo Sapiens
<400> 142
Glu Leu Ser Met His
1 5
<210> 143
<211> 5
<212> PRT
<213> Homo Sapiens
<400> 143
Ser Tyr Gly Met His
1 5
<210> 144
<211> 5
<212> PRT
<213> Homo Sapiens
<400> 144
Ser Tyr Tyr Trp Ser
1 5
<210> 145
<211> 5
<212> PRT
<213> Homo Sapiens
<400> 145
Asn Tyr Tyr Trp Ser
1 5
<210> 146
<211> 5
<212> PRT
<213> Homo Sapiens
<400> 146
Gly Tyr Tyr Ile His
1 5
<210> 147
<211> 5
<212> PRT
<213> Homo Sapiens
<400> 147
Ser Tyr Gly Ile Ser
1 5
<210> 148
<211> 16
<212> PRT
<213> Homo Sapiens
<400> 148
Tyr Ile Tyr Tyr Ser Gly Ser Thr Asn Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 149
<211> 16
<212> PRT
<213> Homo Sapiens
<400> 149
Tyr Ile Ser Tyr Ser Gly Asp Thr Tyr Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 150
<211> 19
<212> PRT
<213> Homo Sapiens
<400> 150
Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala Pro
1 5 10 15
Val Lys Gly
<210> 151
<211> 19
<212> PRT
<213> Homo Sapiens
<400> 151
Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Asn Ala Ala Pro
1 5 10 15
Val Lys Gly
<210> 152
<211> 19
<212> PRT
<213> Homo Sapiens
<400> 152
Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Ala Asp Tyr Ala Ala Pro
1 5 10 15
Val Lys Gly
<210> 153
<211> 19
<212> PRT
<213> Homo Sapiens
<400> 153
Arg Ile Lys Thr Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala Pro
1 5 10 15
Val Lys Gly
<210> 154
<211> 19
<212> PRT
<213> Homo Sapiens
<400> 154
Arg Ile Lys Ser Lys Thr Asp Gly Trp Thr Thr Asp Tyr Ala Ala Pro
1 5 10 15
Val Lys Gly
<210> 155
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 155
Trp Ile Asn Pro Asn Ser Gly Gly Thr Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 156
<211> 16
<212> PRT
<213> Homo Sapiens
<400> 156
Gly Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Pro Gly Ser Val Lys Gly
1 5 10 15
<210> 157
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 157
Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 158
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 158
Val Ile Trp Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 159
<211> 16
<212> PRT
<213> Homo Sapiens
<400> 159
Tyr Ile Tyr Tyr Ser Gly Asn Thr Asn Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 160
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 160
Val Ile Trp Tyr Asp Gly Ser Asn Glu Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 161
<211> 16
<212> PRT
<213> Homo Sapiens
<400> 161
Arg Ile Tyr Thr Ser Gly Ser Thr His Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 162
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 162
Val Ile Trp Tyr Asp Gly Ser Tyr Lys Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 163
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 163
Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu Gln
1 5 10 15
Gly
<210> 164
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 164
Trp Ile Ser Ala Tyr Asn Gly Asn Pro Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 165
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 165
Gly Ile Ala Ala Thr Gly Thr Leu Phe Asp Cys
1 5 10
<210> 166
<211> 15
<212> PRT
<213> Homo Sapiens
<400> 166
Glu Tyr Tyr Gly Ser Gly Gly Val Trp Tyr Tyr Gly Met Asp Val
1 5 10 15
<210> 167
<211> 18
<212> PRT
<213> Homo Sapiens
<400> 167
Gly Gly Ser Leu Leu Trp Thr Gly Pro Asn Tyr Tyr Tyr Tyr Gly Met
1 5 10 15
Asp Val
<210> 168
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 168
Asp Ser Asn Trp Tyr His Asn Trp Phe Asp Pro
1 5 10
<210> 169
<211> 16
<212> PRT
<213> Homo Sapiens
<400> 169
Gly Gly Tyr Ser Gly Tyr Asp Leu Gly Tyr Tyr Tyr Gly Met Asp Val
1 5 10 15
<210> 170
<211> 16
<212> PRT
<213> Homo Sapiens
<400> 170
Glu Gly Pro Tyr Ser Asp Tyr Gly Tyr Tyr Tyr Tyr Gly Met Asp Val
1 5 10 15
<210> 171
<211> 10
<212> PRT
<213> Homo Sapiens
<400> 171
Leu Asp Leu Tyr Gly Asp Tyr Phe Asp Tyr
1 5 10
<210> 172
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 172
Glu Gly Ser Trp Tyr Gly Phe Asp Tyr
1 5
<210> 173
<211> 19
<212> PRT
<213> Homo Sapiens
<400> 173
Glu Tyr Tyr His Ile Leu Thr Gly Ser Phe Tyr Tyr Ser Tyr Tyr Gly
1 5 10 15
Met Asp Val
<210> 174
<211> 19
<212> PRT
<213> Homo Sapiens
<400> 174
Glu Tyr Tyr Gly Ile Val Thr Gly Ser Phe Tyr Tyr Tyr Tyr Tyr Gly
1 5 10 15
Met Asp Val
<210> 175
<211> 15
<212> PRT
<213> Homo Sapiens
<400> 175
Arg Ala Gly Thr Thr Leu Ala Tyr Tyr Tyr Tyr Ala Met Asp Val
1 5 10 15
<210> 176
<211> 10
<212> PRT
<213> Homo Sapiens
<400> 176
Ala Gly Leu Glu Ile Arg Trp Phe Asp Pro
1 5 10
<210> 177
<211> 10
<212> PRT
<213> Homo Sapiens
<400> 177
Ser Ser Gly Asp Tyr Tyr Gly Met Asp Val
1 5 10
<210> 178
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 178
Ile Ala Thr Arg Pro Phe Asp Tyr
1 5
<210> 179
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 179
Asp Arg Val Phe Tyr Gly Met Asp Val
1 5
<210> 180
<211> 12
<212> PRT
<213> Homo Sapiens
<400> 180
Glu Gly Asp Tyr Ser Asp Tyr Tyr Gly Met Asp Val
1 5 10
<210> 181
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 181
Asp Arg Gly Gln Leu Trp Leu Trp Tyr Tyr Tyr Tyr Tyr Gly Met Asp
1 5 10 15
Val
<210> 182
<211> 10
<212> PRT
<213> Homo Sapiens
<400> 182
Ser Ser Gly Asn Tyr Tyr Asp Met Asp Val
1 5 10
<210> 183
<211> 10
<212> PRT
<213> Homo Sapiens
<400> 183
Ser Ser Ser Asn Phe Tyr Asp Met Asp Val
1 5 10
<210> 184
<211> 18
<212> PRT
<213> Homo Sapiens
<400> 184
Asp Leu Arg Ile Thr Gly Thr Thr Tyr Tyr Tyr Tyr Tyr Tyr Gly Met
1 5 10 15
Asp Val
<210> 185
<211> 16
<212> PRT
<213> Homo Sapiens
<400> 185
Asp Gln Gly Leu Leu Gly Phe Gly Glu Leu Glu Gly Leu Phe Asp Tyr
1 5 10 15
<210> 186
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 186
Glu Ser Trp Phe Gly Glu Val Phe Phe Asp Tyr
1 5 10
<210> 187
<211> 20
<212> PRT
<213> Homo Sapiens
<400> 187
Gly Val Met Ile Thr Phe Gly Gly Val Ile Val Gly His Ser Tyr Tyr
1 5 10 15
Gly Met Asp Val
20
<210> 188
<211> 16
<212> PRT
<213> Homo Sapiens
<400> 188
Glu Gly Pro Tyr Ser Asn Tyr Gly Tyr Tyr Tyr Tyr Gly Val Asp Val
1 5 10 15
<210> 189
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 189
Asp Gly Ala Thr Val Val Thr Pro Gly Tyr Tyr Tyr Tyr Gly Thr Asp
1 5 10 15
Val
<210> 190
<211> 10
<212> PRT
<213> Homo Sapiens
<400> 190
Ser Ser Trp Ser Tyr Tyr Gly Met Asp Val
1 5 10
<210> 191
<211> 16
<212> PRT
<213> Homo Sapiens
<400> 191
Arg Ser Ser Gln Ser Leu Val Tyr Ser Asp Gly Asn Thr Tyr Leu Asn
1 5 10 15
<210> 192
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 192
Arg Ala Ser Gln Ser Ile Ser Asp Tyr Leu Asn
1 5 10
<210> 193
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 193
Lys Ser Ser Gln Ser Val Leu Asp Ser Ser Asp Asn Lys Asn Tyr Leu
1 5 10 15
Ala
<210> 194
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 194
Lys Ser Ser Gln Ser Val Leu Asp Ser Ser Asn Asn Lys Asn Phe Leu
1 5 10 15
Ala
<210> 195
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 195
Lys Ser Ser Gln Gly Val Leu Asp Ser Ser Asn Asn Lys Asn Phe Leu
1 5 10 15
Ala
<210> 196
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 196
Arg Ala Ser Gln Ser Ile Ser Arg Tyr Leu Asn
1 5 10
<210> 197
<211> 15
<212> PRT
<213> Homo Sapiens
<400> 197
Lys Ser Ser Gln Ser Leu Leu Ser Asp Gly Lys Thr Tyr Leu Tyr
1 5 10 15
<210> 198
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 198
Gln Ala Ser Gln Asp Ile Ser Asn Tyr Leu Asn
1 5 10
<210> 199
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 199
Gln Ala Ser Gln Asp Ile Asn Asn Tyr Leu Asn
1 5 10
<210> 200
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 200
Gln Ala Ser His Asp Ile Ser Asn Tyr Leu Asn
1 5 10
<210> 201
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 201
Gln Ala Ser Gln Asn Ile Ser Asn Phe Leu Asp
1 5 10
<210> 202
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 202
Gln Ala Ser Gln Asp Ile Ser Asn Phe Leu Asp
1 5 10
<210> 203
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 203
Gln Ala Ser Gln Asp Ile Ser Asn Phe Leu Asn
1 5 10
<210> 204
<211> 17
<212> PRT
<213> Homo Sapiens
<400> 204
Lys Ser Ser Gln Ser Val Leu Tyr Ser Ser Asn Asn Lys Asn Tyr Leu
1 5 10 15
Ala
<210> 205
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 205
Arg Ala Ser Gln Gly Phe Ser Asn Tyr Leu Ala
1 5 10
<210> 206
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 206
Arg Ala Ser Gln Gly Ile Asn Asn Tyr Leu Ala
1 5 10
<210> 207
<211> 16
<212> PRT
<213> Homo Sapiens
<400> 207
Arg Ser Ser Gln Ser Leu Leu His Ser Asn Gly Tyr Asn Tyr Leu Asp
1 5 10 15
<210> 208
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 208
Arg Ala Ser Gln Tyr Ile Gly Ser Ser Leu His
1 5 10
<210> 209
<211> 15
<212> PRT
<213> Homo Sapiens
<400> 209
Arg Ala Ser Gln Ser Val Ser Ser Gly Tyr Leu Ala Tyr Leu Ala
1 5 10 15
<210> 210
<211> 11
<212> PRT
<213> Homo Sapiens
<400> 210
Arg Ala Ser Gln Gly Ile Arg Asn Asp Leu Asp
1 5 10
<210> 211
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 211
Lys Val Ser Asn Trp Asp Ser
1 5
<210> 212
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 212
Ala Ala Ser Ser Leu Gln Ser
1 5
<210> 213
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 213
Trp Ala Ser Thr Arg Glu Ser
1 5
<210> 214
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 214
Trp Ala Ser Asn Arg Glu Ser
1 5
<210> 215
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 215
Glu Ala Ser Asn Arg Phe Ser
1 5
<210> 216
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 216
Asp Ala Ser Asn Leu Glu Thr
1 5
<210> 217
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 217
Asp Thr Ser Asn Leu Glu Pro
1 5
<210> 218
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 218
Asp Ala Ser Asp Leu Asp Pro
1 5
<210> 219
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 219
Asp Ala Ser Asn Leu Glu Ile
1 5
<210> 220
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 220
Ala Ala Ser Thr Leu Gln Ser
1 5
<210> 221
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 221
Val Ala Ser Thr Leu Gln Ser
1 5
<210> 222
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 222
Tyr Val Ser Gln Ser Phe Ser
1 5
<210> 223
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 223
Gly Ala Ser Ser Thr Ala Thr
1 5
<210> 224
<211> 7
<212> PRT
<213> Homo Sapiens
<400> 224
Leu Gly Ser Ile Arg Ala Ser
1 5
<210> 225
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 225
Met Gln Gly Thr His Trp Pro Ile Thr
1 5
<210> 226
<211> 12
<212> PRT
<213> Homo Sapiens
<400> 226
Met Gln Gly Thr His Trp Pro Arg Gly Leu Phe Thr
1 5 10
<210> 227
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 227
Gln Gln Thr Tyr Ser Asp Pro Phe Thr
1 5
<210> 228
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 228
Gln Gln Tyr Tyr Ser Asp Pro Phe Thr
1 5
<210> 229
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 229
Gln Gln Ser Tyr Ile Thr Pro Pro Ser
1 5
<210> 230
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 230
Met Gln Ser Ile Gln Leu Pro Leu Thr
1 5
<210> 231
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 231
Gln Gln Tyr Asp Asn Leu Ile Thr
1 5
<210> 232
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 232
Gln Arg Tyr Asp Asp Leu Pro Ile Thr
1 5
<210> 233
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 233
Gln Gln Tyr Asp Asn Leu Leu Thr
1 5
<210> 234
<211> 8
<212> PRT
<213> Homo Sapiens
<400> 234
Gln Gln Tyr Asp Asp Leu Leu Thr
1 5
<210> 235
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 235
Gln Gln Tyr Asp Asn Leu Pro Leu Thr
1 5
<210> 236
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 236
Gln Gln Tyr Val Ser Leu Pro Leu Thr
1 5
<210> 237
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 237
Gln Gln Tyr Asp Asn Phe Pro Phe Thr
1 5
<210> 238
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 238
Gln Gln Phe Asp Asn Leu Pro Pro Thr
1 5
<210> 239
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 239
Gln Gln Tyr Tyr Thr Thr Pro Pro Thr
1 5
<210> 240
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 240
Gln Gln Tyr Asp Asn Leu Pro Phe Thr
1 5
<210> 241
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 241
Gln Lys Tyr Asn Ser Ala Pro Leu Thr
1 5
<210> 242
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 242
Gln Lys Tyr Asn Ser Gly Pro Phe Thr
1 5
<210> 243
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 243
Met Gln Ala Leu Gln Thr Pro Arg Thr
1 5
<210> 244
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 244
His Gln Ser Ser Ser Leu Pro Phe Thr
1 5
<210> 245
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 245
Gln Gln Tyr Gly Ser Ser Pro Ile Thr
1 5
<210> 246
<211> 9
<212> PRT
<213> Homo Sapiens
<400> 246
Leu Gln Tyr Asn Ser Tyr Pro Ile Thr
1 5
<210> 247
<211> 981
<212> DNA
<213> Homo Sapiens
<400> 247
gctagcacca agggcccatc ggtcttcccc ctggcgccct gctccaggag cacctccgag 60
agcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 120
tggaactcag gcgctctgac cagcggcgtg cacaccttcc cagctgtcct acagtcctca 180
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcaacttcgg cacccagacc 240
tacacctgca acgtagatca caagcccagc aacaccaagg tggacaagac agttgagcgc 300
aaatgttgtg tcgagtgccc accgtgccca gcaccacctg tggcaggacc gtcagtcttc 360
ctcttccccc caaaacccaa ggacaccctc atgatctccc ggacccctga ggtcacgtgc 420
gtggtggtgg acgtgagcca cgaagacccc gaggtccagt tcaactggta cgtggacggc 480
gtggaggtgc ataatgccaa gacaaagcca cgggaggagc agttcaacag cacgttccgt 540
gtggtcagcg tcctcaccgt tgtgcaccag gactggctga acggcaagga gtacaagtgc 600
aaggtctcca acaaaggcct cccagccccc atcgagaaaa ccatctccaa aaccaaaggg 660
cagccccgag aaccacaggt gtacaccctg cccccatccc gggaggagat gaccaagaac 720
caggtcagcc tgacctgcct ggtcaaaggc ttctacccca gcgacatcgc cgtggagtgg 780
gagagcaatg ggcagccgga gaacaactac aagaccacac ctcccatgct ggactccgac 840
ggctccttct tcctctacag caagctcacc gtggacaaga gcaggtggca gcaggggaac 900
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacacgca gaagagcctc 960
tccctgtctc cgggtaaatg a 981
<210> 248
<211> 324
<212> DNA
<213> Homo Sapiens
<400> 248
cgtacggtgg ctgcaccatc tgtcttcatc ttcccgccat ctgatgagca gttgaaatct 60
ggaactgcct ctgttgtgtg cctgctgaat aacttctatc ccagagaggc caaagtacag 120
tggaaggtgg ataacgccct ccaatcgggt aactcccagg agagtgtcac agagcaggac 180
agcaaggaca gcacctacag cctcagcagc accctgacgc tgagcaaagc agactacgag 240
aaacacaaag tctacgcctg cgaagtcacc catcagggcc tgagctcgcc cgtcacaaag 300
agcttcaaca ggggagagtg ttag 324
<210> 249
<211> 375
<212> DNA
<213> Homo Sapiens
<400> 249
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc gcctactata tgcactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcaacccta acagtggtgg cacaaactat 180
gcacagaagt ttcagggcag ggtcaccatg accagggaca cgtccatcag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagaggtgga 300
tatagtggct acgatttggg ctactactac ggtatggacg tctggggcca agggaccacg 360
gtcaccgtct cctca 375
<210> 250
<211> 363
<212> DNA
<213> Homo Sapiens
<400> 250
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcggagac cctgtccctc 60
acctgcactg tctctggtgg ctccgtcagc agtggtggtt actactggag ctggatccgg 120
cagcccccag ggaagggact ggagtggatt gggtatatct attacagtgg gagcaccaac 180
tacaacccct ccctcaagag tcgagtcacc atatcagtag acacgtccaa gaaccagttc 240
tccctgaagc tgagctctgt gaccgctgcg gacacggccg tgtattactg tgcggccggt 300
atagcagcca ctggtaccct ctttgactgc tggggccagg gaaccctggt caccgtctcc 360
tca 363
<210> 251
<211> 378
<212> DNA
<213> Homo Sapiens
<400> 251
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctactata tacactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcaacccta acagtggtgg cacaaactat 180
gcacagaagt ttcagggcag ggtcaccatg accagggaca cgtccatcag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagatcga 300
gggcagctat ggttatggta ctactactac tacggtatgg acgtctgggg ccaagggacc 360
acggtcaccg tctcctca 378
<210> 252
<211> 357
<212> DNA
<213> Homo Sapiens
<400> 252
caggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cgtctggatt caccttcagt agctatggca tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtggcagtt atatggtatg atggaagtaa taaatactat 180
gcagactccg tgaagggccg attcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agccgaggac acggctgtgt attactgtgc cagcagcagc 300
tggtcctact acggtatgga cgtctggggc caagggacca cggtcaccgt ctcctca 357
<210> 253
<211> 387
<212> DNA
<213> Homo Sapiens
<400> 253
gaggtgcagc tggtggagtc tgggggaggc ttggtaaagc ctggggggtc ccttagactc 60
tcctgtgcag cctctggatt cactgtcagt aacgcctgga tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggttggccgt attaaaagca aaactgatgg tgggacaaca 180
gacaacgctg cacccgtgaa aggcagattc accatctcaa gagatgattc aaaaaacacg 240
ctgtatctgc aaatgaacag cctgaaaacc gaggacacag ccgtgtatta ctgtaccaca 300
ggagggtcat tactatggac cgggcccaac tactactact acggtatgga cgtctggggc 360
caagggacca cggtcaccgt ctcctca 387
<210> 254
<211> 378
<212> DNA
<213> Homo Sapiens
<400> 254
gaggtgcagc tggtggagtc tgggggaggc ttggtaaagc ctggggggtc ccttagactc 60
tcctgtgcag cctctggatt cactttcagt aacgcctgga tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggttggccgt attaaaagca aaactgatgg tgggacaaca 180
gactacgctg cacccgtgaa aggcagattc accatctcaa gagatgattc aaaaaacacg 240
ctgtatctgc aaatgaacag cctgaaaacc gaggacacag ccgtgtatta ctgtaccaca 300
gagtactatg gttcgggggg ggtttggtac tacggtatgg acgtctgggg ccaagggacc 360
acggtcaccg tctcctca 378
<210> 255
<211> 387
<212> DNA
<213> Homo Sapiens
<400> 255
gaggtgcagc tggtggagtc tgggggaggc ttggtaaagc ctggggggtc ccttagactc 60
tcctgtgcag cctctggatt cactttcagt aacgcctgga tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggttggccgt attaaaagca aaactgatgg ttggacaaca 180
gactacgctg cacccgtgaa aggcagattc accatctcaa gagatgattc aaaaaacacg 240
ctgtatctgc aaatgaacag cctgaaaacc gaggacacag ccgtgtatta ctgtaccaca 300
gatctccgta taactggaac tacctattac tactactact acggtatgga cgtctggggc 360
caagggacca cggtcaccgt ctcctca 387
<210> 256
<211> 360
<212> DNA
<213> Homo Sapiens
<400> 256
caggttcagc tggtgcagtc tggagctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggtta cacctttacc agctatggta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcagcgctt acaatggtaa cacaaactat 180
gcacagaagc tccagggcag agtcaccatg accacagaca catccacgag cacagcctac 240
atggagctga ggagcctgag atctgacgac acggccgtgt attactgtgc gagagagtcg 300
tggttcgggg aggtattctt tgactactgg ggccagggaa ccctggtcac cgtctcctca 360
<210> 257
<211> 378
<212> DNA
<213> Homo Sapiens
<400> 257
gaggtgcagc tggtggagtc tgggggaggc ttggtaaagc ctggggggtc ccttagactc 60
tcctgtgcag cctctggatt cactttcagt aacgcctgga tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggttggccgt attaaaagca aaactgatgg tgggacaaca 180
gactacgctg cacccgtgaa aggcagattc accatctcaa gagatgattc aaaaaacacg 240
ctgtatctgc aaatgaacag cctgaaaacc gaggacacag ccgtgtatta ctgtaccaca 300
gagtactatg gttcgggggg ggtttggtac tacggtatgg acgtctgggg ccaagggacc 360
acggtcaccg tctcctca 378
<210> 258
<211> 384
<212> DNA
<213> Homo Sapiens
<400> 258
gaggtgcagc tggtggagtc tgggggaggc ttggtaaagc ctggggggtc ccttagactc 60
tcctgtgcag cctctggatt cactttcagt aacgcctgga tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggttggccgt attaaaagca aaactgatgg tgggacaaca 180
gactacgctg cacccgtgaa aggcagattc accatctcaa gagatgattc aaaaaacacg 240
ctgtatctgc aaatgaacag cctgaaaacc gaggacacag ccgtgtatta ctgtaccaca 300
gatggggcta cggtggtaac tccggggtac tactactacg gtacggacgt ctggggccaa 360
gggaccacgg tcaccgtctc ctca 384
<210> 259
<211> 360
<212> DNA
<213> Homo Sapiens
<400> 259
caggttcagc tggtgcagtc tggagctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggtta cacctttacc agctatggta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcagcgctt acaatggtaa cacaaactat 180
gcacagaagc tccagggcag agtcaccatg accacagaca catccacgag cacagcctac 240
atggagctga ggagcctgag atctgacgac acggccgtgt attactgtgc gagagagtcg 300
tggttcgggg aggtattttt tgactactgg ggccagggaa ccctggtcac cgtctcctca 360
<210> 260
<211> 360
<212> DNA
<213> Homo Sapiens
<400> 260
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctactata tgcactgggt gcgacaggcc 120
cctggacaag ggcttgaatg gatgggatgg atcaacccta acagtggtgg cacaaactat 180
gctcagaagt ttcagggcag ggtcaccatg accagggaca cgtccatcag cacagcctac 240
atggagctga gcagactgag atctgacgac acggcctttt attactgtgc gagagacagc 300
aactggtacc acaactggtt cgacccctgg ggccagggaa ccctggtcac cgtctcctca 360
<210> 261
<211> 360
<212> DNA
<213> Homo Sapiens
<400> 261
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctactata tgcactgggt gcgacaggcc 120
cctggacaag ggcttgaatg gatgggatgg atcaacccta acagtggtgg cacaaactat 180
gctcagaagt ttcagggcag ggtcaccatg accagggaca cgtccatcag cacagcctac 240
atggagctga gcagactgag atctgacgac acggcctttt attactgtgc gagagacagc 300
aactggtacc acaactggtt cgacccctgg ggccagggaa ccctggtcac cgtctcctca 360
<210> 262
<211> 384
<212> DNA
<213> Homo Sapiens
<400> 262
gaggtgcagc tggtggagtc tgggggaggc ttggtaaagc ctggggggtc ccttagactc 60
tcctgtgcag cctctggatt cactttcagt aacgcctgga tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggttggccgt attaaaagca aaactgatgg tgggacaaca 180
gactacgctg cacccgtgaa aggcagattc accatctcaa gagatgattc aaaaaacacg 240
ctgtatctgc aaatgaacag cctgaaaacc gaggacacag ccgtgtatta ctgtaccaca 300
gatggggcta cggtggtaac tccggggtac tactactacg gtacggacgt ctggggccaa 360
gggaccacgg tcaccgtctc ctca 384
<210> 263
<211> 381
<212> DNA
<213> Homo Sapiens
<400> 263
gaggtgcaac tggtggagtc tgggggaggc ttggtaaagc ctggggggtc ccttagactc 60
tcctgtgcag cctctggatt cactttcagt aacgcctgga tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggttggccgt attaaaagca aaactgatgg tgggacagca 180
gactacgctg cacccgtgaa aggcagattc accatctcaa gagatgattc aaaaaacacg 240
ctgtatctgc aaatgaacag cctgaaaacc gaggacacag ccgtgtatta ctgtaccaca 300
gaaggtccct acagtgacta cgggtactac tactacggta tggacgtctg gggccaaggg 360
accacggtca ccgtctcctc a 381
<210> 264
<211> 360
<212> DNA
<213> Homo Sapiens
<400> 264
caggttcagc tggtgcagtc tggagctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggtta cacctttacc agctatggta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcagcgctt acaatggtaa cacaaactat 180
gcacagaagc tccagggcag agtcaccatg accacagaca catccacgag cacagcctac 240
atggagctga ggagcctgag atctgacgac acggccgtgt attactgtgc gagagagtcg 300
tggttcgggg aggtattctt tgactactgg ggccagggaa ccctggtcac cgtctcctca 360
<210> 265
<211> 351
<212> DNA
<213> Homo Sapiens
<400> 265
gaggtgcagc tggtggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agctacgaca tgcactgggt ccgccaagct 120
acaggaaaag gtctggagtg ggtctcaggt attggtactg ctggtgacac atactatcca 180
ggctccgtga agggccgatt caacatctcc agagaaaatg ccaagaactc cttgtatctt 240
caaatgaaca gcctgagagc cggggacacg gctgtgtatt actgtgcaag agagggcagc 300
tggtacggct ttgactactg gggccaggga accctggtca ccgtctcctc a 351
<210> 266
<211> 357
<212> DNA
<213> Homo Sapiens
<400> 266
caggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cgtctggatt caccttcagt agctatggca tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtggcagtt atatggtatg atggaagtaa tgaatactat 180
gcagactccg tgaagggccg attcaccatc tccagagaca attccaagag cacgctgtat 240
ctgcaaatga acagcctgag agccgaggac acggctgtgt attactgtgc gcactcgtcc 300
gggaactact acgatatgga cgtctggggc caagggacca cggtcaccgt ctcctca 357
<210> 267
<211> 381
<212> DNA
<213> Homo Sapiens
<400> 267
gaggtgcagc tggtggagtc tgggggaggc ttggtagagc ctggggggtc ccttagactc 60
tcctgtgcag cctctggatt cactttcagt accgcctgga tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggttggccgt attaaaagca aaactgatgg tgggacaaca 180
gactacgctg cacccgtgaa aggcagattc accatctcaa gagatgattc aaaaaacacg 240
ctgtatctgc aaatgaacag cctgaaaaac gaggacacag ccgtgtatta ctgtaccaca 300
gaaggtccct acagtaacta cgggtactac tactacggtg tggacgtctg gggccaaggg 360
accacggtca ccgtctcctc a 381
<210> 268
<211> 357
<212> DNA
<213> Homo Sapiens
<400> 268
caggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cgtctggatt caccttcagt agctatggca tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtggcagtt atatggtatg atggaagtaa taaatactat 180
gcagactccg tgaagggccg attcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agccgaggac acggctgtgt attactgtgc gagcagctcg 300
tcaaacttct acgatatgga cgtctggggc caagggacca cggtcaccgt ctcctca 357
<210> 269
<211> 390
<212> DNA
<213> Homo Sapiens
<400> 269
gaggtgcagc tggtggagtc tgggggaggc ttggtaaagc ctggggggtc ccttacactc 60
tcctgtgcag cctctggatt cactttcaat aacgcctgga tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggttggccgt attaaaagca aaactgatgg tgggacaaca 180
gactacgctg cacccgtgaa aggcagattc accatctcaa gagatgattc aaaaaacacg 240
ctgtatctgc aaatgaacag cctgaaaacc gaggacacag ccgtgtatta ctgtaccaca 300
gaatattacc atattttgac tggttcgttc tactactcct actacggtat ggacgtctgg 360
ggccaaggga ccacggtcac cgtctcctca 390
<210> 270
<211> 351
<212> DNA
<213> Homo Sapiens
<400> 270
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcggagac cctgtccctc 60
acctgcactg tctctggtgg ctccatcagt aattactact ggagctggat ccggcagtcc 120
gccgggaagg gactggagtg gattgggcgt atctatacca gtgggagcac ccactacaac 180
ccctccctca agagtcgaat catcatgtca gtggacacgt ccaagaacca gttctccctg 240
aagctgagct ctgtgaccgc cgcggacacg gccgtgtatt actgtgcgag agatcgagtc 300
ttctacggta tggacgtctg gggccaaggg accacggtca ccgtctcctc a 351
<210> 271
<211> 363
<212> DNA
<213> Homo Sapiens
<400> 271
caggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cgtctggatt caccttcagt agctatggca tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtggcagtt atatggtatg atggaagtta taaatactat 180
gcagactccg tgaagggccg attcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agccgaggac acggctgtgt attactgtgc gagagaaggg 300
gattactccg actactacgg tatggacgtc tggggccaag ggaccacggt caccgtctcc 360
tca 363
<210> 272
<211> 387
<212> DNA
<213> Homo Sapiens
<400> 272
caggtccagc tggtacagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg tttccggata caccctcact gaattatcca tgcactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gatgggaggt tttgatcctg aagatggtga aacaatctac 180
gcacagaagt tccagggcag agtcaccatg accgaggaca catctacaga cacagtttac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc aacaggggtt 300
atgattacgt ttgggggagt tatcgttggc cactcctact acggtatgga cgtctggggc 360
caagggacca cggtcaccgt ctcctca 387
<210> 273
<211> 372
<212> DNA
<213> Homo Sapiens
<400> 273
caggtccagc tggtacagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg tttccggata caccctcact gaattatcca tgcactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gatgggaggt tttgatcctg aagatggtga aacaatctac 180
gcacagaagt tccagggcag agtcaccatg accgaggaca catctacaga cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc aacaagggct 300
ggaacgacgt tggcctacta ctactacgct atggacgtct ggggccaagg gaccacggtc 360
accgtctcct ca 372
<210> 274
<211> 348
<212> DNA
<213> Homo Sapiens
<400> 274
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcggagac cctgtccctc 60
acctgcactg tctctggtgg ctccatcagt agttactact ggagctggat ccggcagccc 120
ccagggaagg gactggagtg gattgggtat atctattaca gtgggaacac caactacaac 180
ccctccctca agagtcgatt caccttatca atagacacgt ccaagaacca gttctccctg 240
aggctgagct ctgtgaccgc tgcggacacg gccgtgtatt actgtgcgtg tatagcaact 300
cggccctttg actactgggg ccagggaacc ctggtcaccg tctcctca 348
<210> 275
<211> 357
<212> DNA
<213> Homo Sapiens
<400> 275
caggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cgtcaggatt caccttcatc agctatggca tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtggcagtt atatggtatg atggaagtaa taaatactat 180
gcagactccg tgaagggccg attcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agccgaggac acggctgtgt attactgtgc ggatagcagt 300
ggcgactact acggtatgga cgtctggggc caagggacca cggtcaccgt ctcctca 357
<210> 276
<211> 357
<212> DNA
<213> Homo Sapiens
<400> 276
caggtccagc tggtacagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg tttccggata caccctcact gaattatcca tgcactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gatgggaggt tttgatcctg aagatggtga aacaatctac 180
gcacagaagt tccagggcag agtcaccatg accgaggaca catctacaga cacagcctat 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc aacagcgggg 300
ctggaaatac ggtggttcga cccctggggc cagggaaccc tggtcaccgt ctcctca 357
<210> 277
<211> 360
<212> DNA
<213> Homo Sapiens
<400> 277
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcacagac cctgtccctc 60
acctgcactg tctctggtgg ctccatcagc agtggtggtt actactggag ctggatccgc 120
cagcacccag ggaagggcct ggagtggatt gggtacatct cttacagtgg ggacacctac 180
tacaacccgt ccctcaagag tcgacttacc atatcagtag acacgtctaa gcaccagttc 240
tccctgaggc tgagctctgt gacttccgcg gacacggccg tgtattactg tgcgagtcta 300
gacctctacg gtgactactt tgactactgg ggccagggaa ccctggtcac cgtctcctca 360
<210> 278
<211> 375
<212> DNA
<213> Homo Sapiens
<400> 278
caggttcagc tggtgcagtc tggagctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggtta caccttaacc agctatggta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcagcgctt acaatggtaa cccaaactat 180
gcacagaagt tccagggcag agtcaccatg accacagaca catccacgag cacagcctac 240
atggagctga ggagcctgag atctgacgac acggccgtgt attactgtgc gagagatcag 300
ggattactag ggttcgggga actcgagggg ctctttgact actggggcca gggaaccctg 360
gtcaccgtct cctca 375
<210> 279
<211> 390
<212> DNA
<213> Homo Sapiens
<400> 279
gaggtgcagc tggtggagtc tgggggaggc ttggtaaagc ctggggggtc ccttagactc 60
tcctgtgcag cctctggatt cactttcagt aacgcctgga tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggttggccgt attaaaacca aaactgatgg tgggacaaca 180
gactacgctg cacccgtgaa aggcagattc accatctcaa gagatgattc acaaaacacg 240
ctgtatctgc aaatgaacag cctgaaaacc gaggacacag ccgtgtatta ctgtaccaca 300
gaatattacg gtattgtgac tggttcgttt tattactact actacggtat ggacgtctgg 360
ggccaaggga ccacggtcac cgtctcctca 390
<210> 280
<211> 321
<212> DNA
<213> Homo Sapiens
<400> 280
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc aggcgagtca aaacattagc aactttttag attggtatca gcagaaacca 120
gggaaagccc ctaacctcct gatctacgat gcatccgatt tggatccagg ggtcccatca 180
aggttcagtg gaagtggatc tgggacagat tttactttca ccatcagcag cctacagcct 240
gaagatattg caacatatta ctgtcaacag tatgttagtc tcccgctcac tttcggcgga 300
gggaccaagg tggagatcaa a 321
<210> 281
<211> 336
<212> DNA
<213> Homo Sapiens
<400> 281
gataatgtga tgacccagac tccactctct ctgtccgtca cccctggaca gccggcctcc 60
atctcctgca agtcgagtca gagcctcctg catagtgatg ggaagaccta tttgtattgg 120
tacctgcaga agccaggcca gcctccacag ctcctgatct atgaagcttc caaccggttc 180
tctggagtgc cagataggtt cagtggcagc gggtcaggga cagatttcac actgaaaatc 240
agccgggtgg aggctgagga tgttggggtt tattactgca tgcaaagtat acagcttcct 300
ctcactttcg gcggagggac caaggtggag atcaaa 336
<210> 282
<211> 321
<212> DNA
<213> Homo Sapiens
<400> 282
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc aggcgagtca ggacattaac aactatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctacgat gcatccaatt tggaaatagg ggtcccatca 180
aggttcagtg gaagtggatc tgggacagat ttcattttca ccatcagcag tctgcagcct 240
gaagatattg caacatatta ctgtcaacag tatgataatt tcccgttcac tttcggcgga 300
gggaccaagg tggagatcaa a 321
<210> 283
<211> 318
<212> DNA
<213> Homo Sapiens
<400> 283
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc aggcgagtca ggacattaac aactatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctacgat acatccaatt tggaaccagg ggtcccatca 180
aggttcagtg gaagtggatc tgggacagat tttactttca ccatcagcag cctgcagcct 240
gaagatattg caacatatta ctgtcaacaa tatgataatc tcctcacctt cggccaaggg 300
acacgactgg aaattaaa 318
<210> 284
<211> 318
<212> DNA
<213> Homo Sapiens
<400> 284
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc aggcgagtca ggacattagc aactatttaa attggtatca gcataaacca 120
gggaaagccc ctaaactcct gatctacgat gcatccaatt tggaaacagg ggtcccatca 180
aggttcagtg gaagtggatc tgggacagat tttactttca ccatcagcag cctgcagcct 240
gaagatattg caacatatta ctgtcaacag tatgataatc tgctcacctt cggcggaggg 300
accaaggtgg agatcaaa 318
<210> 285
<211> 318
<212> DNA
<213> Homo Sapiens
<400> 285
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc aggcgagtca ggacattagc aactatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagttcct gatctacgat gcatccaatt tggaaacagg ggtcccatca 180
aggtttagtg gaagtggatc tgggacagat tttactttca ccatcagcag cctgcagcct 240
gaagatattg caacatatta ctgtcaacag tatgataatc tgatcacctt cggccaaggg 300
acacgactgg agattaaa 318
<210> 286
<211> 339
<212> DNA
<213> Homo Sapiens
<400> 286
gacatcgtga tgacccagtc tccagactcc ctggctgtgt ctctgggcga gagggccacc 60
atcaactgca agtccagcca gagtgtttta gacagctccg acaataagaa ctacttagct 120
tggtaccagc agaaaccagg acagcctcct aagctgctca tttactgggc atctaaccgg 180
gaatccgggg tccctgaccg attcagtggc agcgggtctg ggacagattt ctctctcacc 240
atcagcagcc tgcaggctga agatgtggca gtttattact gtcagcaata ttatagtgat 300
ccattcactt tcggccctgg gaccaaagtg gatatcaaa 339
<210> 287
<211> 318
<212> DNA
<213> Homo Sapiens
<400> 287
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggtga cagagtcacc 60
atcacttgcc aggcgagtca ggacattagc aactatttaa attggtatca acagaaacca 120
gggaaagccc ctaaactcct gatctacgat gcatccaatt tggaaacagg ggtcccatca 180
aggttcagtg gaagtggatc tgggacagat tttactttca ccatcagcag cctacagcct 240
gaagatattg caacatatta ctgtcaacag tatgataatc tgctcacttt cggcggaggg 300
accaaggtgg agatcaaa 318
<210> 288
<211> 339
<212> DNA
<213> Homo Sapiens
<400> 288
gacatcgtga tgacccagtc tccagactcc ctggctgtgt ctctgggcga gagggccacc 60
atcgactgca agtccagcca gggtgtttta gacagctcca acaataagaa cttcttagct 120
tggtaccagc agaaaccagg acagcctcct aagctgctca tttactgggc atctaaccgg 180
gaatccgggg tccctgtccg attcagtggc agcgggtctg ggacagattt cactctcacc 240
atcagcagcc tgcaggctga agatgtggca ctttattact gtcagcaata ttatagtgat 300
ccattcactt tcggccctgg gaccaaagtg gatatcaaa 339
<210> 289
<211> 321
<212> DNA
<213> Homo Sapiens
<400> 289
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagt gactatttaa attggtatca gcagaaacca 120
gggaaagccc ctaacctcct gatctatgct gcatccagtt tgcagagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttactt ctgtcaacag acttacagtg acccattcac tttcggccct 300
gggaccaaag tggatatcaa a 321
<210> 290
<211> 318
<212> DNA
<213> Homo Sapiens
<400> 290
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc aggcgagtca ggacattagc aactatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctacgat gcatccaatt tggaaacagg ggtcccatca 180
aggttcagtg gaagtggatc tgggacagat tttactttca ccatcagcag cctacagcct 240
gaagatattg caacatatta ctgtcaacag tatgataatc tgctcacttt cggcggaggg 300
accaaggtgg agatcaaa 318
<210> 291
<211> 318
<212> DNA
<213> Homo Sapiens
<400> 291
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc aggcgagtca ggacattagc aactatttaa attggtatca gcagaaacca 120
gggaaagccc ctaaggtcct gatctacgat gcatccaatt tggaaacagg ggtcccatca 180
aggttcagtg gaagtggatc tgggacagat tttactttca ccatcagcag cctgcagcct 240
gaagatattg caacatatta ctgtcaacag tatgataatc tcctcacttt cggcggaggg 300
accaaggtgg agatcaaa 318
<210> 292
<211> 339
<212> DNA
<213> Homo Sapiens
<400> 292
gacatcgtga tgacccagtc tccagactcc ctggctgtgt ctctgggcga gagggccacc 60
atcgactgca agtccagcca gagtgtttta gacagctcca acaataagaa cttcttagct 120
tggtaccagc agaaaccagg acagcctcct aagctgctca tttactgggc atctaaccgg 180
gaatccgggg tccctgaccg attcagtggc agcgggtctg ggacagattt cactctcacc 240
atcagcagcc tgcaggctga agatgtggca gtttattact gtcagcaata ttatagtgat 300
ccattcactt tcggccctgg gaccaaagtg gatatcaaa 339
<210> 293
<211> 333
<212> DNA
<213> Homo Sapiens
<400> 293
gaaattgtgt tgacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agcggctact tagcctactt agcctggtac 120
cagcagaaac ctggccaggc tcccaggctc ctcatctatg gtgcatccag cacggccact 180
ggcatcccag acaggttcag tggcagtggg tctgggacag acttcactct caccatcagc 240
agactggagc ctgaagattt tgcagtgtat tactgtcagc agtatggtag ctcaccgatc 300
accttcggcc aagggacacg actggagatt aaa 333
<210> 294
<211> 321
<212> DNA
<213> Homo Sapiens
<400> 294
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc aggcgagtca ggacattagc aactttttaa attggtatca gcagagacca 120
gggaaagccc ctaagctcct gatctacgat gcatccaatt tggaaacagg ggtcccatca 180
aggttcagtg gaagtggatc tgggacagat tttactttca ccatcagcag cctgcagcct 240
gaagatattg caacatatta ctgtcaacag tatgataatc tcccattcac tttcggccct 300
gggaccaaag tggatatcaa a 321
<210> 295
<211> 318
<212> DNA
<213> Homo Sapiens
<400> 295
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc aggcgagtca ggacattagc aactatttaa attggtatca gcagaaacca 120
gggaaagccc ctaaactcct gatctacgat gcatccaatt tggaaacagg ggtcccatca 180
aggttcagtg gaagtggatc tgggacagat tttactttca ccatcagcag cctgcagcct 240
gaagattttg caacatatta ctgtcaacag tatgataatc tcctcacttt cggcggaggg 300
accaaggtgg agatcaaa 318
<210> 296
<211> 321
<212> DNA
<213> Homo Sapiens
<400> 296
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc aggcgagtca ggacattagc aactatttaa attggtatca gcagaaacca 120
ggaaaagccc ctaagctcct gatctacgat gcatccaatt tggaaacagg ggtcccatca 180
aggttcagtg gaagtggatc tgggacagat tttactttca ccatcagcag cctgcagcct 240
gaagatattg caacatatta ctgtcaacgg tatgatgatc tcccgatcac cttcggccaa 300
gggacacgac tggagattaa a 321
<210> 297
<211> 318
<212> DNA
<213> Homo Sapiens
<400> 297
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtgggaga cagagtcacc 60
atcacttgcc aggcgagtca ggacattagc aactatttaa attggtatca gcagagacca 120
gggaaagccc ctaagctcct gatctacgat gcatccaatt tggaaacagg ggtcccatca 180
aggttcagtg gaagtggatc tgggacagat tttactttca ccatcagcag cctgcagcct 240
gaagatattg caacatatta ctgtcaacag tatgataatc tgctcacttt cggcggaggg 300
accaaggtgg agatcaaa 318
<210> 298
<211> 321
<212> DNA
<213> Homo Sapiens
<400> 298
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcgagtca gggctttagc aattatttag cctggtatca gcagaaacca 120
gggaaagttc ctaagctcct gatctatgct gcatccactt tgcagtcagg ggtcccatct 180
cggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagatgttg caacttatta ctgtcaaaag tataacagtg ccccgctcac tttcggcgga 300
gggaccaagg tggagatcaa a 321
<210> 299
<211> 321
<212> DNA
<213> Homo Sapiens
<400> 299
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcgagtca gggcattaac aattatttag cctggtatca gcagaaacca 120
gggaaagttc ctcagctcct gatctatgtt gcatccactt tgcaatcagg ggtcccatct 180
cggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagatgttg caacttatta ctgtcaaaag tataacagtg gcccattcac tttcggccct 300
gggaccaaag tggatatcaa a 321
<210> 300
<211> 321
<212> DNA
<213> Homo Sapiens
<400> 300
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc aggtatttaa attggtatca gcagaaacca 120
gggaaagccc ctaacctcct gatccatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcaacag agttacatta cccctcccag ttttggccag 300
gggaccaagc tggagatcaa a 321
<210> 301
<211> 321
<212> DNA
<213> Homo Sapiens
<400> 301
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gggcattaga aatgatttag actggtatca gcagaaacca 120
gggaaagccc ctaagcgcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatct 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca caatcaacag cctgcagcct 240
gaagattttg caacttatta ctgtctacag tataatagtt acccgatcac cttcggccaa 300
gggacacgac tggagattaa a 321
<210> 302
<211> 321
<212> DNA
<213> Homo Sapiens
<400> 302
gacatccaga tgatccagtc tccttcctcc ctgtctgcat ctgtcggaga cagagtcacc 60
atcacttgcc aggcgagtca cgacattagc aactatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagttcct gatctccgat gcatccaatt tggaaacagg ggtcccatca 180
aggttcagtg gaagtggatc tgggacagat tttactttca ccatcagcag cctgcagcct 240
gaagatattg caacatatta ctgtcaacag tatgataatc tcccgctcac tttcggcgga 300
gggaccaagg tggagatcaa a 321
<210> 303
<211> 336
<212> DNA
<213> Homo Sapiens
<400> 303
gatattgtga tgactcagtc tccactctcc ctgcccgtca cccctggaga gccggcctcc 60
atctcctgca ggtctagtca gagcctcctg catagtaatg gatacaacta tttggattgg 120
tacctgcaga agccagggca gtcaccacag ttcctgatct atttgggttc tattcgggcc 180
tccggggtcc ctgacaggtt cagtggcagt ggatcaggca cagattttgc actgacaatc 240
agcagagtgg aggctgagga tgttggggtt tattactgca tgcaagctct acaaactcct 300
cggacgttcg gccaagggac caaggtggaa atcaaa 336
<210> 304
<211> 321
<212> DNA
<213> Homo Sapiens
<400> 304
gaaattgtgc tgactcagtc tccagacttt cagtctgtga ctccaaagga gaaagtcacc 60
atcacctgcc gggccagtca gtacattggt agtagcttac actggtacca gcagacacca 120
gatcagtctc caaagctcct catcaactat gtttcccagt ccttctcagg ggtcccctcg 180
aggttcagtg gcagtggatc tgggacagat ttcaccctca ccatcaatag cctggaagct 240
gaagatgctg caacgtatta ctgtcatcag agtagtagtt taccattcac tttcggccct 300
gggaccaaag tggatatcaa a 321
<210> 305
<211> 339
<212> DNA
<213> Homo Sapiens
<400> 305
gacatcgtga tgacccagtc tccagactcc ctggctgtgt ctctgggcgc gagggccacc 60
atctcctgca agtccagcca gagtgtttta tacagctcca acaataagaa ctacttagct 120
tggtaccagc agaaaccagg ccagcctcct aagctgctca tttactgggc atctacccgg 180
gaatccgggg tccctgaccg attcagtggc agcgggtctg ggacagattt cactctcacc 240
atcagcaccc tgcaggctga agatgtggca gtttattact gtcagcaata ttatactact 300
cctccgacgt tcggccaagg gaccaaggtg gaaatcaaa 339
<210> 306
<211> 318
<212> DNA
<213> Homo Sapiens
<400> 306
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc aggcgagtca ggacattaac aactatttaa attggtatca acagaaacca 120
gggaaagccc ctaaactcct gatctacgat gcatccaatt tggaaacagg ggtcccatca 180
aggttcagtg gaagtggatc tgggacagat tttactttca ccatcagcag cctgcagcct 240
gaagatattg caacatatta ctgtcaacag tatgatgatc tgctcacttt cggcggaggg 300
accaaggtgg agatcaaa 318
<210> 307
<211> 10
<212> PRT
<213> Homo Sapiens
<220>
<221> VARIANT
<222> 4
<223> Xaa = Phe or Leu
<400> 307
Gly Tyr Thr Xaa Thr Ser Tyr Gly Ile Ser
1 5 10
<210> 308
<211> 17
<212> PRT
<213> Homo Sapiens
<220>
<221> VARIANT
<222> 9
<223> Xaa = Thr or Pro
<220>
<221> VARIANT
<222> 15
<223> Xaa = Leu or Phe
<400> 308
Trp Ile Ser Ala Tyr Asn Gly Asn Xaa Asn Tyr Ala Gln Lys Xaa Gln
1 5 10 15
Gly
<210> 309
<211> 16
<212> PRT
<213> Homo Sapiens
<220>
<221> VARIANT
<222> 1
<223> Xaa = Glu or Asp
<220>
<221> VARIANT
<222> 2
<223> Xaa = Ser or Gln
<220>
<221> VARIANT
<222> 3
<223> Xaa = Gly or none
<220>
<221> VARIANT
<222> 4, 5
<223> Xaa = Leu or none
<220>
<221> VARIANT
<222> 6
<223> Xaa = Trp or Gly
<220>
<221> VARIANT
<222> 10
<223> Xaa = Val or Leu
<220>
<221> VARIANT
<222> (11)...(11)
<223> Xaa = Glu or none
<220>
<221> VARIANT
<222> (12)...(12)
<223> Xaa = Gly or none
<220>
<221> VARIANT
<222> (13)...(13)
<223> Xaa = Phe or Leu
<400> 309
Xaa Xaa Xaa Xaa Xaa Xaa Phe Gly Glu Xaa Xaa Xaa Xaa Phe Asp Tyr
1 5 10 15
<210> 310
<211> 17
<212> PRT
<213> Homo Sapiens
<220>
<221> VARIANT
<222> 4
<223> Xaa = Gln or Ser
<220>
<221> VARIANT
<222> 8
<223> Xaa = Asp or Tyr
<220>
<221> VARIANT
<222> 11
<223> Xaa = Asn or Asp
<220>
<221> VARIANT
<222> 15
<223> Xaa = Phe or Tyr
<400> 310
Lys Ser Ser Xaa Gly Val Leu Xaa Ser Ser Xaa Asn Lys Asn Xaa Leu
1 5 10 15
Ala
<210> 311
<211> 7
<212> PRT
<213> Homo Sapiens
<220>
<221> VARIANT
<222> 4
<223> Xaa = Asn or Thr
<400> 311
Trp Ala Ser Xaa Arg Glu Ser
1 5
<210> 312
<211> 9
<212> PRT
<213> Homo Sapiens
<220>
<221> VARIANT
<222> 5
<223> Xaa = Ser or Thr
<220>
<221> VARIANT
<222> 6
<223> Xaa = Asp or Thr
<220>
<221> VARIANT
<222> 8
<223> Xaa = Phe or Pro
<400> 312
Gln Gln Tyr Tyr Xaa Xaa Pro Xaa Thr
1 5
<210> 313
<211> 10
<212> PRT
<213> Homo Sapiens
<220>
<221> VARIANT
<222> 4
<223> Xaa = Phe or Val
<220>
<221> VARIANT
<222> 5
<223> Xaa = Ser or Asn
<220>
<221> VARIANT
<222> 6
<223> Xaa = Asn or Thr
<400> 313
Gly Phe Thr Xaa Xaa Xaa Ala Trp Met Ser
1 5 10
<210> 314
<211> 19
<212> PRT
<213> Homo Sapiens
<220>
<221> VARIANT
<222> 4
<223> Xaa = Ser or Thr
<220>
<221> VARIANT
<222> 9
<223> Xaa = Gly or Trp
<220>
<221> VARIANT
<222> 11
<223> Xaa = Thr or Ala
<220>
<221> VARIANT
<222> 13
<223> Xaa = Tyr or Asn
<400> 314
Arg Ile Lys Xaa Lys Thr Asp Gly Xaa Thr Xaa Asp Xaa Ala Ala Pro
1 5 10 15
Val Lys Gly
<210> 315
<211> 19
<212> PRT
<213> Homo Sapiens
<220>
<221> VARIANT
<222> 1
<223> Xaa = Glu, Asp or Gly
<220>
<221> VARIANT
<222> 2
<223> Xaa = Tyr, Leu or none
<220>
<221> VARIANT
<222> 3
<223> Xaa = Tyr, Arg or Gly
<220>
<221> VARIANT
<222> 4
<223> Xaa = His, Gly, Ser, or none
<220>
<221> VARIANT
<222> 5
<223> Xaa = Ile, Ala, Leu, or none
<220>
<221> VARIANT
<222> 6
<223> Xaa = Leu, Val, Tyr, Pro or none
<220>
<221> VARIANT
<222> (7)...(7)
<223> Xaa = Thr, Val, Tyr, Gly, Trp, or none
<220>
<221> VARIANT
<222> (8)...(8)
<223> Xaa = Gly, Val, Ser, or Thr
<220>
<221> VARIANT
<222> (9)...(9)
<223> Xaa = Ser, Thr, Asp, Asn, or Gly
<220>
<221> VARIANT
<222> (10)...(10)
<223> Xaa = Gly, Phe, Pro, or Tyr
<220>
<221> VARIANT
<222> (11)...(11)
<223> Xaa = Gly, Tyr, or Asn
<220>
<221> VARIANT
<222> (12)...(12)
<223> Xaa = Val or Tyr
<220>
<221> VARIANT
<222> (13)...(13)
<223> Xaa = Trp, Ser or Tyr
<220>
<221> VARIANT
<222> (17)...(17)
<223> Xaa = Met, Thr, or Val
<400> 315
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Tyr Tyr Gly
1 5 10 15
Xaa Asp Val
<210> 316
<211> 11
<212> PRT
<213> Homo Sapiens
<220>
<221> VARIANT
<222> 7
<223> Xaa = Ser or Asn
<400> 316
Gln Ala Ser Gln Asp Ile Xaa Asn Tyr Leu Asn
1 5 10
<210> 317
<211> 7
<212> PRT
<213> Homo Sapiens
<220>
<221> VARIANT
<222> 2
<223> Xaa = Ala or Thr
<220>
<221> VARIANT
<222> 7
<223> Xaa = Thr or Pro
<400> 317
Asp Xaa Ser Asn Leu Glu Xaa
1 5
<210> 318
<211> 8
<212> PRT
<213> Homo Sapiens
<220>
<221> VARIANT
<222> 5
<223> Xaa = Asn or Asp
<220>
<221> VARIANT
<222> 7
<223> Xaa = Leu or Ile
<400> 318
Gln Gln Tyr Asp Xaa Leu Xaa Thr
1 5
<210> 319
<211> 10
<212> PRT
<213> Homo Sapiens
<220>
<221> VARIANT
<222> 5
<223> Xaa = Ser or Ile
<400> 319
Gly Phe Thr Phe Xaa Ser Tyr Gly Met His
1 5 10
<210> 320
<211> 17
<212> PRT
<213> Homo Sapiens
<220>
<221> VARIANT
<222> 9
<223> Xaa = Glu or Lys
<400> 320
Val Ile Trp Tyr Asp Gly Ser Asn Xaa Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 321
<211> 10
<212> PRT
<213> Homo Sapiens
<220>
<221> VARIANT
<222> 3
<223> Xaa = Gly, Ser or Trp
<220>
<221> VARIANT
<222> 4
<223> Xaa = Asn, Asp or Ser
<220>
<221> VARIANT
<222> 5
<223> Xaa = Tyr or Phe
<220>
<221> VARIANT
<222> 7
<223> Xaa = Asp or Gly
<400> 321
Ser Ser Xaa Xaa Xaa Tyr Xaa Met Asp Val
1 5 10
<210> 322
<211> 11
<212> PRT
<213> Homo Sapiens
<220>
<221> VARIANT
<222> 4
<223> Xaa = Gln or His
<220>
<221> VARIANT
<222> 7
<223> Xaa = Ser or Asn
<220>
<221> VARIANT
<222> 9
<223> Xaa = Phe or Tyr
<400> 322
Gln Ala Ser Xaa Asp Ile Xaa Asn Xaa Leu Asn
1 5 10
<210> 323
<211> 7
<212> PRT
<213> Homo Sapiens
<220>
<221> VARIANT
<222> 7
<223> Xaa = Thr or Ile
<400> 323
Asp Ala Ser Asn Leu Glu Xaa
1 5
<210> 324
<211> 9
<212> PRT
<213> Homo Sapiens
<220>
<221> VARIANT
<222> 2
<223> Xaa = Gln or Arg
<220>
<221> VARIANT
<222> 5
<223> Xaa = Asn or Asp
<220>
<221> VARIANT
<222> 6
<223> Xaa = Leu or Phe
<220>
<221> VARIANT
<222> 8
<223> Xaa = Phe, Leu or Ile
<400> 324
Gln Xaa Tyr Asp Xaa Xaa Pro Xaa Thr
1 5
<210> 325
<211> 12
<212> DNA
<213> Homo Sapiens
<400> 325
gggaaaggga aa 12
<210> 326
<211> 203
<212> PRT
<213> Homo Sapiens
<400> 326
Ile Pro Val Ile Glu Pro Ser Val Pro Glu Leu Val Val Lys Pro Gly
1 5 10 15
Ala Thr Val Thr Leu Arg Cys Val Gly Asn Gly Ser Val Glu Trp Asp
20 25 30
Gly Pro Pro Ser Pro His Trp Thr Leu Tyr Ser Asp Gly Ser Ser Ser
35 40 45
Ile Leu Ser Thr Asn Asn Ala Thr Phe Gln Asn Thr Gly Thr Tyr Arg
50 55 60
Cys Thr Glu Pro Gly Asp Pro Leu Gly Gly Ser Ala Ala Ile His Leu
65 70 75 80
Tyr Val Lys Asp Pro Ala Arg Pro Trp Asn Val Leu Ala Gln Glu Val
85 90 95
Val Val Phe Glu Asp Gln Asp Ala Leu Leu Pro Cys Leu Leu Thr Asp
100 105 110
Pro Val Leu Glu Ala Gly Val Ser Leu Val Arg Val Arg Gly Arg Pro
115 120 125
Leu Met Arg His Thr Asn Tyr Ser Phe Ser Pro Trp His Gly Phe Thr
130 135 140
Ile His Arg Ala Lys Phe Ile Gln Ser Gln Asp Tyr Gln Cys Ser Ala
145 150 155 160
Leu Met Gly Gly Arg Lys Val Met Ser Ile Ser Ile Arg Leu Lys Val
165 170 175
Gln Lys Val Ile Pro Gly Pro Pro Ala Leu Thr Leu Val Pro Ala Leu
180 185 190
Val Arg Ile Arg Gly Glu Ala Ala Gln Ile Val
195 200
Claims (67)
- 중쇄 가변영역(VH) 및 경쇄 가변영역(VL)을 포함하며, 여기에서:
VH 및 VL이 이하에서 규정되는 바와 같은 아미노산 즉:
(a) VH가 SEQ ID NO:77을 포함하고 그리고 VL이 SEQ ID NO:109를 포함하거나;
(b) VH가 SEQ ID NO:77을 포함하고 그리고 VL이 SEQ ID NO:11O을 포함하거나;
(c) VH가 SEQ ID NO:78을 포함하고 그리고 VL이 SEQ ID NO:133을 포함하거나;
(d) VH가 SEQ ID NO:79를 포함하고 그리고 VL이 SEQ ID NO:111을 포함하거나;
(e) VH가 SEQ ID NO:80을 포함하고 그리고 VL이 SEQ ID NO:112를 포함하거나;
(f) VH가 SEQ ID NO:84를 포함하고 그리고 VL이 SEQ ID NO:115를 포함하거나;
(g) VH가 SEQ ID NO:85를 포함하고 그리고 VL이 SEQ ID NO:116을 포함하거나;
(h) VH가 SEQ ID NO:86을 포함하고 그리고 VL이 SEQ ID NO:117을 포함하거나;
(i) VH가 SEQ ID NO:87을 포함하고 그리고 VL이 SEQ ID NO:118을 포함하거나;
(j) VH가 SEQ ID NO:70을 포함하고 그리고 VL이 SEQ ID NO:102를 포함하거나;
(k) VH가 SEQ ID NO:70을 포함하고 그리고 VL이 SEQ ID NO:103을 포함하거나;
(l) VH가 SEQ ID NO:73을 포함하고 그리고 VL이 SEQ ID NO:105를 포함하거나;
(m) VH가 SEQ ID NO:74를 포함하고 그리고 VL이 SEQ ID NO:106을 포함하거나;
(n) VH가 SEQ ID NO:89를 포함하고 그리고 VL이 SEQ ID NO:121을 포함하거나;
(o) VH가 SEQ ID NO:93을 포함하고 그리고 VL이 SEQ ID NO:123을 포함하거나;
(p) VH가 SEQ ID NO:94를 포함하고 그리고 VL이 SEQ ID NO:124를 포함하거나;
(q) VH가 SEQ ID NO:97을 포함하고 그리고 VL이 SEQ ID NO:127을 포함하거나;
(r) VH가 SEQ ID NO:98을 포함하고 그리고 VL이 SEQ ID NO:128을 포함하거나; 또는
(s) VH가 SEQ ID NO:99를 포함하고 그리고 VL이 SEQ ID NO:129를 포함하는 것을 특징으로 하는 c-fms와 결합하는 항원 결합 단백질. - 제 1 항에 있어서,
상기 항원 결합 단백질이 상보적 결정 영역들(CDRs) CDRH1, CDRH2, CDRH3, CDRL1, CDRL2 및 CDRL3를 포함하며, 여기에서
상기 CDRH1이 시퀀스 GYTx1TSYGIS(SEQ ID NO:307)를 포함하고, 여기에서 x1은 F 및 L로 이루어지는 그룹으로부터 선택되는 것이고;
상기 CDRH2가 시퀀스 WISAYNGNX1NYAQKX2QG(SEQ ID NO:308)를 포함하고, 여기에서 X1은 T 및 P로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X2는 L 및 F로 이루어지는 그룹으로부터 선택되는 것이고;
상기 CDRH3이 시퀀스 X1X2X3X4X5FGEX6X7X8X9FDY(SEQ ID NO:309)를 포함하고, 여기에서 X1은 E 및 D로 이루어지는 그룹으로부터 선택되는 것이고, X2는 S 및 Q로 이루어지는 그룹으로부터 선택되는 것이고, X3은 G 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X4는 L 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X5는 W 및 G로 이루어지는 그룹으로부터 선택되는 것이고, X6은 V 및 L로 이루어지는 그룹으로부터 선택되는 것이고, X7은 E 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X8은 G 및 무 아미노산으로 이루어지는 그룹으로부터 선택되는 것이고, X9는 F 및 L로 이루어지는 그룹으로부터 선택되는 것이고;
상기 CDRL1이 시퀀스 KSSX1GVLX2SSX3NKNX4LA(SEQ ID NO:310)를 포함하고, 여기에서 X1은 Q 및 S로 이루어지는 그룹으로부터 선택되는 것이고, X2는 D 및 Y로 이루어지는 그룹으로부터 선택되는 것이고, X3은 N 및 D로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X4는 F 및 Y로 이루어지는 그룹으로부터 선택되는 것이고;
상기 CDRL2는 시퀀스 WASX1RES(SEQ ID NO:311)를 포함하고, 여기에서 X1은 N 및 T로 이루어지는 그룹으로부터 선택되는 것이고; 그리고
상기 CDRL3는 시퀀스 QQYYX1X2PX3T(SEQ ID NO:312)를 포함하고, 여기에서 X1은 S 및 T로 이루어지는 그룹으로부터 선택되는 것이고, X2는 D 및 T로 이루어지는 그룹으로부터 선택되는 것이고, 그리고 X3은 F 및 P로 이루어지는 그룹으로부터 선택되는 것임을 특징으로 하는 상기 항원 결합 단백질. - 제 1 항에 있어서,
상기 항원 결합 단백질이 상보적 결정 영역들(CDRs) CDRH1, CDRH2, CDRH3, CDRL1, CDRL2 및 CDRL3를 포함하며, 여기에서 상기 CDRs들이 이하에서 규정되는 바와 같은 아미노산 시퀀스들 즉:
(a) CDRH1이 SEQ ID NO:147을 포함하고, CDRH2가 SEQ ID NO:163을 포함하고, CDRH3이 SEQ ID NO:186을 포함하고, CDRL1이 SEQ ID NO:193을 포함하고, CDRL2가 SEQ ID NO:214를 포함하고, 그리고 CDRL3이 SEQ ID NO:228을 포함하고;
(b) CDRH1이 SEQ ID NO:137을 포함하고, CDRH2가 SEQ ID NO:150을 포함하고, CDRH3이 SEQ ID NO:166을 포함하고, CDRL1이 SEQ ID NO:198을 포함하고, CDRL2가 SEQ ID NO:216을 포함하고, 그리고 CDRL3이 SEQ ID NO:233을 포함하고;
(c) CDRH1이 SEQ ID NO:137을 포함하고, CDRH2가 SEQ ID NO:150을 포함하고, CDRH3이 SEQ ID NO:189를 포함하고, CDRL1이 SEQ ID NO:198을 포함하고, CDRL2가 SEQ ID NO:216을 포함하고, 그리고 CDRL3이 SEQ ID NO:233을 포함하고;
(d) CDRH1이 SEQ ID NO:147을 포함하고, CDRH2가 SEQ ID NO:163을 포함하고, CDRH3이 SEQ ID NO:186을 포함하고, CDRL1이 SEQ ID NO:195를 포함하고, CDRL2가 SEQ ID NO:214를 포함하고, 그리고 CDRL3이 SEQ ID NO:228을 포함하고;
(e) CDRH1이 SEQ ID NO:137을 포함하고, CDRH2가 SEQ ID NO:152를 포함하고, CDRH3이 SEQ ID NO:170을 포함하고, CDRL1이 SEQ ID NO:198을 포함하고, CDRL2가 SEQ ID NO:216을 포함하고, 그리고 CDRL3이 SEQ ID NO:233을 포함하고;
(f) CDRH1이 SEQ ID NO:147을 포함하고, CDRH2가 SEQ ID NO:163을 포함하고, CDRH3이 SEQ ID NO:186을 포함하고, CDRL1이 SEQ ID NO:194를 포함하고, CDRL2가 SEQ ID NO:214를 포함하고, 그리고 CDRL3이 SEQ ID NO:228을 포함하고;
(g) CDRH1이 SEQ ID NO:141을 포함하고, CDRH2가 SEQ ID NO:156을 포함하고, CDRH3이 SEQ ID NO:172를 포함하고, CDRL1이 SEQ ID NO:209를 포함하고, CDRL2가 SEQ ID NO:223을 포함하고, 그리고 CDRL3이 SEQ ID NO:245를 포함하고;
(h) CDRH1이 SEQ ID NO:143을 포함하고, CDRH2가 SEQ ID NO:160을 포함하고, CDRH3이 SEQ ID NO:182를 포함하고, CDRL1이 SEQ ID NO:203을 포함하고, CDRL2가 SEQ ID NO:216을 포함하고, 그리고 CDRL3이 SEQ ID NO:240을 포함하고;
(i) CDRH1이 SEQ ID NO:140을 포함하고, CDRH2가 SEQ ID NO:155를 포함하고, CDRH3이 SEQ ID NO:169를 포함하고, CDRL1이 SEQ ID NO:202를 포함하고, CDRL2가 SEQ ID NO:218을 포함하고, 그리고 CDRL3이 SEQ ID NO:236을 포함하고;
(j) CDRH1이 SEQ ID NO:140을 포함하고, CDRH2가 SEQ ID NO:155를 포함하고, CDRH3이 SEQ ID NO:169를 포함하고, CDRL1이 SEQ ID NO:201을 포함하고, CDRL2가 SEQ ID NO:218을 포함하고, 그리고 CDRL3이 SEQ ID NO:236을 포함하고;
(k) CDRH1이 SEQ ID NO:143을 포함하고, CDRH2가 SEQ ID NO:158을 포함하고, CDRH3이 SEQ ID NO:190을 포함하고, CDRL1이 SEQ ID NO:199를 포함하고, CDRL2가 SEQ ID NO:219를 포함하고, 그리고 CDRL3이 SEQ ID NO:237을 포함하고;
(l) CDRH1이 SEQ ID NO:137을 포함하고, CDRH2가 SEQ ID NO:151을 포함하고, CDRH3이 SEQ ID NO:167을 포함하고, CDRL1이 SEQ ID NO:199를 포함하고, CDRL2가 SEQ ID NO:217을 포함하고, 그리고 CDRL3이 SEQ ID NO:233을 포함하고;
(m) CDRH1이 SEQ ID NO:137을 포함하고, CDRH2가 SEQ ID NO:150을 포함하고, CDRH3이 SEQ ID NO:173을 포함하고, CDRL1이 SEQ ID NO:198을 포함하고, CDRL2가 SEQ ID NO:216을 포함하고, 그리고 CDRL3이 SEQ ID NO:233을 포함하고;
(n) CDRH1이 SEQ ID NO:142를 포함하고, CDRH2가 SEQ ID NO:157을 포함하고, CDRH3이 SEQ ID NO:187을 포함하고, CDRL1이 SEQ ID NO:206을 포함하고, CDRL2가 SEQ ID NO:221을 포함하고, 그리고 CDRL3이 SEQ ID NO:242를 포함하고;
(o) CDRH1이 SEQ ID NO:143을 포함하고, CDRH2가 SEQ ID NO:158을 포함하고, CDRH3이 SEQ ID NO:177을 포함하고, CDRL1이 SEQ ID NO:200을 포함하고, CDRL2가 SEQ ID NO:216을 포함하고, 그리고 CDRL3이 SEQ ID NO:235를 포함하고;
(p) CDRH1이 SEQ ID NO:142를 포함하고, CDRH2가 SEQ ID NO:157을 포함하고, CDRH3이 SEQ ID NO:176을 포함하고, CDRL1이 SEQ ID NO:207을 포함하고, CDRL2가 SEQ ID NO:224를 포함하고, 그리고 CDRL3이 SEQ ID NO:243을 포함하고; 그리고
(q) CDRH1이 SEQ ID NO:136을 포함하고, CDRH2가 SEQ ID NO:149를 포함하고, CDRH3이 SEQ ID NO:171을 포함하고, CDRL1이 SEQ ID NO:208을 포함하고, CDRL2가 SEQ ID NO:222를 포함하고, 그리고 CDRL3이 SEQ ID NO:244를 포함하는 것을 특징으로 하는 상기 항원 결합 단백질. - 삭제
- 제 1 항에 있어서,
상기 항원 결합 단백질이 완전 중쇄 및 완전 경쇄를 포함하고, 여기에서 상기 완전 중쇄 및 완전 경쇄가 이하에서 규정되는 바와 같은 아미노산 즉:
(a) 상기 완전 중쇄가 SEQ ID NO:11을 포함하고 그리고 상기 완전 경쇄가 SEQ ID NO:43을 포함하거나;
(b) 상기 완전 중쇄가 SEQ ID NO:11을 포함하고 그리고 상기 완전 경쇄가 SEQ ID NO:44를 포함하거나;
(c) 상기 완전 중쇄가 SEQ ID NO:12를 포함하고 그리고 상기 완전 경쇄가 SEQ ID NO:67을 포함하거나;
(d) 상기 완전 중쇄가 SEQ ID NO:13을 포함하고 그리고 상기 완전 경쇄가 SEQ ID NO:45를 포함하거나;
(e) 상기 완전 중쇄가 SEQ ID NO:14를 포함하고 그리고 상기 완전 경쇄가 SEQ ID NO:46을 포함하거나;
(f) 상기 완전 중쇄가 SEQ ID NO:18을 포함하고 그리고 상기 완전 경쇄가 SEQ ID NO:49를 포함하거나;
(g) 상기 완전 중쇄가 SEQ ID NO:19를 포함하고 그리고 상기 완전 경쇄가 SEQ ID NO:50을 포함하거나;
(h) 상기 완전 중쇄가 SEQ ID NO:20을 포함하고 그리고 상기 완전 경쇄가 SEQ ID NO:51을 포함하거나;
(i) 상기 완전 중쇄가 SEQ ID NO:21을 포함하고 그리고 상기 완전 경쇄가 SEQ ID NO:52를 포함하거나;
(j) 상기 완전 중쇄가 SEQ ID NO:4를 포함하고 그리고 상기 완전 경쇄가 SEQ ID NO:36을 포함하거나;
(k) 상기 완전 중쇄가 SEQ ID NO:4를 포함하고 그리고 상기 완전 경쇄가 SEQ ID NO:37을 포함하거나;
(l) 상기 완전 중쇄가 SEQ ID NO:7을 포함하고 그리고 상기 완전 경쇄가 SEQ ID NO:39를 포함하거나;
(m) 상기 완전 중쇄가 SEQ ID NO:8을 포함하고 그리고 상기 완전 경쇄가 SEQ ID NO:40을 포함하거나;
(n) 상기 완전 중쇄가 SEQ ID NO:23을 포함하고 그리고 상기 완전 경쇄가 SEQ ID NO:55를 포함하거나;
(o) 상기 완전 중쇄가 SEQ ID NO:27을 포함하고 그리고 상기 완전 경쇄가 SEQ ID NO:57을 포함하거나;
(p) 상기 완전 중쇄가 SEQ ID NO:28을 포함하고 그리고 상기 완전 경쇄가 SEQ ID NO:58을 포함하거나;
(q) 상기 완전 중쇄가 SEQ ID NO:31을 포함하고 그리고 상기 완전 경쇄가 SEQ ID NO:61을 포함하거나;
(r) 상기 완전 중쇄가 SEQ ID NO:32를 포함하고 그리고 상기 완전 경쇄가 SEQ ID NO:62를 포함하거나; 또는
(s) 상기 완전 중쇄가 SEQ ID NO:33을 포함하고 그리고 상기 완전 경쇄가 SEQ ID NO:63을 포함하는 것을 특징으로 하는 상기 항원 결합 단백질. - 삭제
- 삭제
- 제 1 항에 있어서,
상기 항원 결합 단백질은 모노클로날 항체, 재조합 항체, 인간 항체, 인간화된 항체, 키메라 항체, 이종특이성 항체 또는 다중특이성 항체이거나;
IgG1, IgG2, IgG3 또는 IgG4 아형(subtype)이거나;
IgG1, IgG2, IgG3 또는 IgG4 아형의 인간 항체이거나; 또는
Fab 단편, Fab' 단편, F(ab')2 단편, an Fv 단편, 다이어바디, 도메인 항체 또는 단쇄 항체 분자인 것을 특징으로 하는 상기 항원 결합 단백질. - 제 1 항에 따른 상기 항원 결합 단백질을 인코딩하고, 선택적으로 여기에서 핵산이 조절시퀀스에 작동가능하게 연결되는 것을 특징으로 하는 핵산.
- 제 9 항에 따른 상기 핵산을 포함하는 벡터.
- 제 9 항에 따른 상기 핵산 또는 제 10 항에 따른 상기 벡터를 포함하는 숙주세포.
- 제 1 항에 따른 적어도 하나의 항원 결합 단백질 및 약제학적으로 수용가능한 부형제를 포함하는 종양 억제용 약제학적 조성물.
- 제 12 항에 있어서,
항염증제, 암요법제, 골성장촉진제(동화작용제) 및 골 항-재흡수제로 이루어지는 그룹으로부터 선택되는 활성제를 더 포함하는 것을 특징으로 하는 상기 종양 억제용 약제학적 조성물. - 치료에 사용하기 위한 제 1 항에 따른 상기 항원 결합 단백질.
- 암, 골질환 및 염증성 질환으로부터 선택되는 상태를 치료하거나 또는 예방하는 데 사용하기 위한 제 1 항에 따른 상기 항원 결합 단백질.
- 제 14 항에 있어서,
상기 항원 결합 단백질이 항-염증제, 암요법제, 골성장촉진제(동화작용제) 또는 골 항-재흡수제와 함께 투여되는 것을 특징으로 하는 상기 항원 결합 단백질. - 제 11 항에 따른 숙주세포를 배양하여 항원 결합 단백질을 생산하는 단계를 포함하는 것을 특징으로 하는 제 1 항에 따른 상기 항원 결합 단백질을 제조하는 방법.
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
- 삭제
Applications Claiming Priority (5)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US95714807P | 2007-08-21 | 2007-08-21 | |
| US60/957,148 | 2007-08-21 | ||
| US8458808P | 2008-07-29 | 2008-07-29 | |
| US61/084,588 | 2008-07-29 | ||
| PCT/US2008/073611 WO2009026303A1 (en) | 2007-08-21 | 2008-08-19 | Human c-fms antigen binding proteins |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| KR20100056492A KR20100056492A (ko) | 2010-05-27 |
| KR101770429B1 true KR101770429B1 (ko) | 2017-08-22 |
Family
ID=40019258
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020107005166A KR101770429B1 (ko) | 2007-08-21 | 2008-08-19 | 인간 c-fms 항원 결합 단백질 |
Country Status (30)
| Country | Link |
|---|---|
| US (4) | US8182813B2 (ko) |
| EP (4) | EP3330292A1 (ko) |
| JP (3) | JP5718640B2 (ko) |
| KR (1) | KR101770429B1 (ko) |
| CN (1) | CN101802008B (ko) |
| AR (1) | AR068347A1 (ko) |
| AU (1) | AU2008288974B2 (ko) |
| BR (1) | BRPI0815368A2 (ko) |
| CA (1) | CA2696761C (ko) |
| CL (1) | CL2008002444A1 (ko) |
| CR (1) | CR11282A (ko) |
| CY (1) | CY1119755T1 (ko) |
| DK (1) | DK2188313T3 (ko) |
| EA (2) | EA023555B1 (ko) |
| ES (1) | ES2650224T3 (ko) |
| HR (1) | HRP20171741T1 (ko) |
| HU (1) | HUE037265T2 (ko) |
| LT (1) | LT2188313T (ko) |
| MX (1) | MX2010001918A (ko) |
| NO (1) | NO2188313T3 (ko) |
| NZ (1) | NZ583282A (ko) |
| PE (1) | PE20091004A1 (ko) |
| PL (1) | PL2188313T3 (ko) |
| PT (1) | PT2188313T (ko) |
| RS (1) | RS56743B1 (ko) |
| SG (1) | SG10201500328QA (ko) |
| SI (1) | SI2188313T1 (ko) |
| TW (1) | TWI595005B (ko) |
| WO (1) | WO2009026303A1 (ko) |
| ZA (1) | ZA201001541B (ko) |
Families Citing this family (81)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US8470977B2 (en) | 2008-03-14 | 2013-06-25 | Transgene S.A. | Antibody against the CSF-1R |
| CN102702358B (zh) | 2008-03-14 | 2016-02-17 | 天士力创世杰(天津)生物制药有限公司 | 针对csf-1r的抗体 |
| US8080246B2 (en) | 2008-11-26 | 2011-12-20 | Five Prime Therapeutics, Inc. | Colony stimulating factor 1 receptor (CSF1R) extracellular domain fusion molecules |
| US8183207B2 (en) | 2008-11-26 | 2012-05-22 | Five Prime Therapeutics, Inc. | Treatment of osteolytic disorders and cancer using CSF1R extracellular domain fusion molecules |
| IT1394281B1 (it) * | 2009-01-19 | 2012-06-06 | Zardi | Processo per la produzione di proteine di fusione polivalenti e polispecifiche utilizzando come struttura portante l'uteroglobina e prodotti cosi' ottenuti. |
| US20110002945A1 (en) * | 2009-07-01 | 2011-01-06 | O'nuallain Brian | Use of immunoglobulin heavy and light chains or fragments thereof to bind to aggregated amyloidogenic proteins |
| PT2949670T (pt) | 2009-12-10 | 2019-05-20 | Hoffmann La Roche | Anticorpos que se ligam preferencialmente ao domínio extracelular 4 do csf1r e respetiva utilização |
| KR101656548B1 (ko) | 2010-03-05 | 2016-09-09 | 에프. 호프만-라 로슈 아게 | 인간 csf-1r에 대한 항체 및 이의 용도 |
| BR112012022046A2 (pt) * | 2010-03-05 | 2017-02-14 | F Hoffamann-La Roche Ag | ''anticorpo,composição farmacêutica,ácido nucleico ,vetores de expressão,célula hospedeira e método para a produção de um anticorpo recombinante''. |
| AR080698A1 (es) * | 2010-04-01 | 2012-05-02 | Imclone Llc | Anticuerpo o fragmento del mismo que especificamente enlaza la variante de csf -1r humano, composicion farmaceutica que lo comprende, su uso para la manufactura de un medicamento util para el tratamiento de cancer y metodo para determinar si un sujeto es candidato para tratamiento de cancer basado e |
| SI2566517T1 (sl) | 2010-05-04 | 2019-01-31 | Five Prime Therapeutics, Inc. | Protitelesa, ki vežejo CSF1R |
| EA031044B1 (ru) * | 2010-10-13 | 2018-11-30 | Янссен Байотек, Инк. | Человеческие антитела к онкостатину м и способы их применения |
| CN103443285B9 (zh) | 2011-02-08 | 2018-04-24 | 米迪缪尼有限公司 | 特异性结合金黄色葡萄球菌α毒素的抗体以及使用方法 |
| CN102719404B (zh) * | 2011-03-30 | 2015-02-18 | 中国人民解放军军事医学科学院毒物药物研究所 | 用于筛选c-Fms激酶抑制剂的细胞模型及筛选方法 |
| JP6323718B2 (ja) | 2011-05-17 | 2018-05-16 | ザ ロックフェラー ユニバーシティー | ヒト免疫不全ウイルスを無毒化する抗体、およびその使用方法。 |
| JP2014520873A (ja) | 2011-07-18 | 2014-08-25 | ザ ユニバーシティ オブ メルボルン | c−Fmsアンタゴニストの使用 |
| CA2851771C (en) | 2011-10-21 | 2018-09-11 | Transgene Sa | Modulation of macrophage activation |
| US10023643B2 (en) | 2011-12-15 | 2018-07-17 | Hoffmann-La Roche Inc. | Antibodies against human CSF-1R and uses thereof |
| AR090263A1 (es) | 2012-03-08 | 2014-10-29 | Hoffmann La Roche | Terapia combinada de anticuerpos contra el csf-1r humano y las utilizaciones de la misma |
| US20130302322A1 (en) | 2012-05-11 | 2013-11-14 | Five Prime Therapeutics, Inc. | Methods of treating conditions with antibodies that bind colony stimulating factor 1 receptor (csf1r) |
| SG10201906328RA (en) | 2012-08-31 | 2019-08-27 | Five Prime Therapeutics Inc | Methods of treating conditions with antibodies that bind colony stimulating factor 1 receptor (csf1r) |
| US9708375B2 (en) | 2013-03-15 | 2017-07-18 | Amgen Inc. | Inhibitory polypeptides specific to WNT inhibitors |
| KR20150140752A (ko) | 2013-04-12 | 2015-12-16 | 모르포시스 아게 | M-csf를 표적으로 하는 항체 |
| AR095882A1 (es) * | 2013-04-22 | 2015-11-18 | Hoffmann La Roche | Terapia de combinación de anticuerpos contra csf-1r humano con un agonista de tlr9 |
| GB201315487D0 (en) * | 2013-08-30 | 2013-10-16 | Ucb Pharma Sa | Antibodies |
| GB201315486D0 (en) * | 2013-08-30 | 2013-10-16 | Ucb Pharma Sa | Antibodies |
| EP3041863A4 (en) | 2013-09-05 | 2017-08-16 | Amgen Inc. | Fc-containing molecules exhibiting predictable, consistent, and reproducible glycoform profiles |
| AR097584A1 (es) | 2013-09-12 | 2016-03-23 | Hoffmann La Roche | Terapia de combinación de anticuerpos contra el csf-1r humano y anticuerpos contra el pd-l1 humano |
| MX2016014761A (es) | 2014-05-16 | 2017-05-25 | Amgen Inc | Ensayo para detectar poblaciones celulares linfocitos t colaboradores 1 (th1) y linfocitos t colaboradores (th2). |
| WO2015200089A1 (en) | 2014-06-23 | 2015-12-30 | Five Prime Therapeutics, Inc. | Methods of treating conditions with antibodies that bind colony stimulating factor 1 receptor (csf1r) |
| WO2016069727A1 (en) | 2014-10-29 | 2016-05-06 | Five Prime Therapeutics, Inc. | Combination therapy for cancer |
| BR112017013111A2 (pt) | 2014-12-22 | 2018-05-15 | Five Prime Therapeutics Inc | métodos de tratamento de um distúrbio e de tratamento da sinovite, uso de um anticorpo e anticorpo |
| BR112017020952A2 (pt) | 2015-04-13 | 2018-07-10 | Five Prime Therapeutics Inc | método de tratamento de câncer, composição e uso da composição |
| US20180147271A1 (en) * | 2015-05-18 | 2018-05-31 | Bluebird Bio, Inc. | Anti-ror1 chimeric antigen receptors |
| WO2016189045A1 (en) | 2015-05-27 | 2016-12-01 | Ucb Biopharma Sprl | Method for the treatment of neurological disease |
| EP3108897A1 (en) | 2015-06-24 | 2016-12-28 | F. Hoffmann-La Roche AG | Antibodies against human csf-1r for use in inducing lymphocytosis in lymphomas or leukemias |
| PE20181322A1 (es) | 2015-09-01 | 2018-08-14 | Agenus Inc | Anticuerpo anti-pd1 y sus metodos de uso |
| KR20180054701A (ko) * | 2015-09-16 | 2018-05-24 | 아블렉시스, 엘엘씨 | 항-cd115 항체 |
| EA201892362A1 (ru) | 2016-04-18 | 2019-04-30 | Селлдекс Терапьютикс, Инк. | Агонистические антитела, которые связываются с cd40 человека, и варианты их применения |
| CN109475603B (zh) | 2016-06-20 | 2023-06-13 | 科马布有限公司 | 抗pd-l1抗体 |
| CN109790220A (zh) | 2016-08-25 | 2019-05-21 | 豪夫迈·罗氏有限公司 | 与巨噬细胞激活剂组合的抗csf-1r抗体的间歇给药 |
| CN110072553B (zh) | 2016-12-22 | 2023-09-15 | 豪夫迈·罗氏有限公司 | 在抗pd-l1/pd1治疗失败之后抗csf-1r抗体与抗pd-l1抗体组合对肿瘤的治疗 |
| WO2018183366A1 (en) | 2017-03-28 | 2018-10-04 | Syndax Pharmaceuticals, Inc. | Combination therapies of csf-1r or csf-1 antibodies and a t-cell engaging therapy |
| JP2020512357A (ja) | 2017-03-31 | 2020-04-23 | ファイブ プライム セラピューティクス, インコーポレイテッド | 抗gitr抗体を使用した癌の併用療法 |
| AU2018256406A1 (en) | 2017-04-19 | 2019-10-17 | Marengo Therapeutics, Inc. | Multispecific molecules and uses thereof |
| CN111278459A (zh) | 2017-05-19 | 2020-06-12 | 赛达克斯制药股份有限公司 | 组合疗法 |
| JP7164141B2 (ja) | 2017-06-08 | 2022-11-01 | 株式会社フリーハンド | 帽子 |
| MA49950A (fr) | 2017-08-25 | 2020-07-01 | Five Prime Therapeutics Inc | Anticorps anti-b7-h4 et leurs procédés d'utilisation |
| BR112020004879A2 (pt) | 2017-09-13 | 2020-09-15 | Five Prime Therapeutics, Inc. | métodos para tratar o câncer pancreático, para tratar o câncer e para determinar a responsividade de um sujeito com câncer |
| WO2019113464A1 (en) | 2017-12-08 | 2019-06-13 | Elstar Therapeutics, Inc. | Multispecific molecules and uses thereof |
| WO2019169212A1 (en) | 2018-03-02 | 2019-09-06 | Five Prime Therapeutics, Inc. | B7-h4 antibodies and methods of use thereof |
| CA3093853A1 (en) | 2018-03-26 | 2019-10-03 | Amgen Inc. | Total afucosylated glycoforms of antibodies produced in cell culture |
| EA202092464A1 (ru) | 2018-04-12 | 2021-01-27 | Эмджен Инк. | Способы получения стабильных композиций на основе белков |
| CA3108282A1 (en) | 2018-08-02 | 2020-02-06 | Dyne Therapeutics, Inc. | Muscle targeting complexes and uses thereof for treating dystrophinopathies |
| CN113164777A (zh) | 2018-09-27 | 2021-07-23 | 马伦戈治疗公司 | Csf1r/ccr2多特异性抗体 |
| CA3115633A1 (en) | 2018-10-09 | 2020-04-16 | Medimmune, Llc | Combinations of anti-staphylococcus aureus antibodies |
| WO2020124039A1 (en) * | 2018-12-13 | 2020-06-18 | Development Center For Biotechnology | Anti-human csf-1r antibody and uses thereof |
| BR112021016791A2 (pt) * | 2019-02-26 | 2021-11-16 | Sorrento Therapeutics Inc | Proteínas de ligação de antígenos que se ligam a bcma |
| CA3140914A1 (en) * | 2019-05-24 | 2020-12-03 | Elixiron Immunotherapeutics (hong Kong) Limited | Anti-csf1r antibodies, il10 fusion proteins, and uses thereof |
| US20220233691A1 (en) | 2019-05-30 | 2022-07-28 | Bristol-Myers Squibb Company | Cell localization signature and combination therapy |
| CN114174538A (zh) | 2019-05-30 | 2022-03-11 | 百时美施贵宝公司 | 适合于免疫肿瘤学疗法的多肿瘤基因特征 |
| EP3976832A1 (en) | 2019-05-30 | 2022-04-06 | Bristol-Myers Squibb Company | Methods of identifying a subject suitable for an immuno-oncology (i-o) therapy |
| JP2022547718A (ja) * | 2019-09-13 | 2022-11-15 | メモリアル スローン ケタリング キャンサー センター | 抗cd371抗体およびその使用 |
| WO2021055817A1 (en) * | 2019-09-19 | 2021-03-25 | New York Society For The Ruptured And Crippled Maintaining The Hospital For Special Surgery | Stabilized c-fms intracellular fragments (ficd) promote osteoclast differentiation and arthritic bone erosion |
| CN114430747A (zh) | 2019-09-26 | 2022-05-03 | 豪夫迈·罗氏有限公司 | 抗csf-1r抗体 |
| US20220349898A1 (en) | 2019-09-26 | 2022-11-03 | Amgen Inc. | Methods of producing antibody compositions |
| CN111068054B (zh) * | 2019-11-06 | 2022-02-11 | 浙江大学医学院附属第一医院 | 以csf1r作为药物靶点治疗肿瘤的药剂及其制备方法 |
| US20230273126A1 (en) | 2020-06-04 | 2023-08-31 | Amgen Inc. | Assessment of cleaning procedures of a biotherapeutic manufacturing process |
| MX2023002326A (es) | 2020-08-31 | 2023-03-21 | Bristol Myers Squibb Co | Firma de localizacion celular e inmunoterapia. |
| US20240043501A1 (en) | 2020-10-15 | 2024-02-08 | Amgen Inc. | Relative unpaired glycans in antibody production methods |
| WO2022120179A1 (en) | 2020-12-03 | 2022-06-09 | Bristol-Myers Squibb Company | Multi-tumor gene signatures and uses thereof |
| JP2023552812A (ja) * | 2020-12-09 | 2023-12-19 | ジャナックス セラピューティクス,インク. | Psmaおよびエフェクタ細胞抗原を標的とする腫瘍活性化抗体に関連する組成物ならびに方法 |
| EP4065161A1 (en) | 2020-12-14 | 2022-10-05 | Ammax Bio, Inc | High concentration formulations of anti-csf1 and anti-csf1r antibodies |
| TW202317614A (zh) | 2021-06-07 | 2023-05-01 | 美商安進公司 | 使用岩藻糖苷酶控制糖基化蛋白的去岩藻糖基化水平 |
| IL309936A (en) * | 2021-07-09 | 2024-03-01 | Dyne Therapeutics Inc | Muscle targeting complexes and formulations for the treatment of dystrophinopathy |
| US11771776B2 (en) | 2021-07-09 | 2023-10-03 | Dyne Therapeutics, Inc. | Muscle targeting complexes and uses thereof for treating dystrophinopathies |
| CN113712526B (zh) * | 2021-09-30 | 2022-12-30 | 四川大学 | 一种脉搏波提取方法、装置、电子设备及存储介质 |
| CA3233279A1 (en) | 2021-10-05 | 2023-04-13 | Amgen, Inc. | Fc-gamma receptor ii binding and glycan content |
| WO2023178329A1 (en) | 2022-03-18 | 2023-09-21 | Bristol-Myers Squibb Company | Methods of isolating polypeptides |
| US11931421B2 (en) | 2022-04-15 | 2024-03-19 | Dyne Therapeutics, Inc. | Muscle targeting complexes and formulations for treating myotonic dystrophy |
| WO2023215725A1 (en) | 2022-05-02 | 2023-11-09 | Fred Hutchinson Cancer Center | Compositions and methods for cellular immunotherapy |
Citations (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2004045532A2 (en) | 2002-11-15 | 2004-06-03 | Chiron Corporation | Methods for preventing and treating cancer metastasis and bone loss associated with cancer metastasis |
Family Cites Families (123)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US3773919A (en) | 1969-10-23 | 1973-11-20 | Du Pont | Polylactide-drug mixtures |
| US4263428A (en) | 1978-03-24 | 1981-04-21 | The Regents Of The University Of California | Bis-anthracycline nucleic acid function inhibitors and improved method for administering the same |
| US4399216A (en) | 1980-02-25 | 1983-08-16 | The Trustees Of Columbia University | Processes for inserting DNA into eucaryotic cells and for producing proteinaceous materials |
| IE52535B1 (en) | 1981-02-16 | 1987-12-09 | Ici Plc | Continuous release pharmaceutical compositions |
| DE3374837D1 (en) | 1982-02-17 | 1988-01-21 | Ciba Geigy Ag | Lipids in the aqueous phase |
| US4816567A (en) | 1983-04-08 | 1989-03-28 | Genentech, Inc. | Recombinant immunoglobin preparations |
| HUT35524A (en) | 1983-08-02 | 1985-07-29 | Hoechst Ag | Process for preparing pharmaceutical compositions containing regulatory /regulative/ peptides providing for the retarded release of the active substance |
| US4615885A (en) | 1983-11-01 | 1986-10-07 | Terumo Kabushiki Kaisha | Pharmaceutical composition containing urokinase |
| US4740461A (en) | 1983-12-27 | 1988-04-26 | Genetics Institute, Inc. | Vectors and methods for transformation of eucaryotic cells |
| US4751180A (en) | 1985-03-28 | 1988-06-14 | Chiron Corporation | Expression using fused genes providing for protein product |
| US4935233A (en) | 1985-12-02 | 1990-06-19 | G. D. Searle And Company | Covalently linked polypeptide cell modulators |
| EP0272253A4 (en) | 1986-03-07 | 1990-02-05 | Massachusetts Inst Technology | METHOD FOR IMPROVING GLYCOPROTE INSTABILITY. |
| US4959455A (en) | 1986-07-14 | 1990-09-25 | Genetics Institute, Inc. | Primate hematopoietic growth factors IL-3 and pharmaceutical compositions |
| DE3785186T2 (de) | 1986-09-02 | 1993-07-15 | Enzon Lab Inc | Bindungsmolekuele mit einzelpolypeptidkette. |
| US5260203A (en) | 1986-09-02 | 1993-11-09 | Enzon, Inc. | Single polypeptide chain binding molecules |
| US4946778A (en) | 1987-09-21 | 1990-08-07 | Genex Corporation | Single polypeptide chain binding molecules |
| US4912040A (en) | 1986-11-14 | 1990-03-27 | Genetics Institute, Inc. | Eucaryotic expression system |
| US5011912A (en) | 1986-12-19 | 1991-04-30 | Immunex Corporation | Hybridoma and monoclonal antibody for use in an immunoaffinity purification system |
| US4965195A (en) | 1987-10-26 | 1990-10-23 | Immunex Corp. | Interleukin-7 |
| US4968607A (en) | 1987-11-25 | 1990-11-06 | Immunex Corporation | Interleukin-1 receptors |
| KR970002917B1 (ko) | 1988-05-27 | 1997-03-12 | 씨네겐 인코포레이티드 | 인터루킨-i 억제제 |
| GB8823869D0 (en) | 1988-10-12 | 1988-11-16 | Medical Res Council | Production of antibodies |
| WO1990005183A1 (en) | 1988-10-31 | 1990-05-17 | Immunex Corporation | Interleukin-4 receptors |
| US5530101A (en) | 1988-12-28 | 1996-06-25 | Protein Design Labs, Inc. | Humanized immunoglobulins |
| JP2762522B2 (ja) | 1989-03-06 | 1998-06-04 | 藤沢薬品工業株式会社 | 血管新生阻害剤 |
| US5112946A (en) | 1989-07-06 | 1992-05-12 | Repligen Corporation | Modified pf4 compositions and methods of use |
| US5683888A (en) | 1989-07-22 | 1997-11-04 | University Of Wales College Of Medicine | Modified bioluminescent proteins and their use |
| US5292658A (en) | 1989-12-29 | 1994-03-08 | University Of Georgia Research Foundation, Inc. Boyd Graduate Studies Research Center | Cloning and expressions of Renilla luciferase |
| JP3068180B2 (ja) | 1990-01-12 | 2000-07-24 | アブジェニックス インコーポレイテッド | 異種抗体の生成 |
| US6673986B1 (en) | 1990-01-12 | 2004-01-06 | Abgenix, Inc. | Generation of xenogeneic antibodies |
| US6713610B1 (en) | 1990-01-12 | 2004-03-30 | Raju Kucherlapati | Human antibodies derived from immunized xenomice |
| WO1991018982A1 (en) | 1990-06-05 | 1991-12-12 | Immunex Corporation | Type ii interleukin-1 receptors |
| ATE158021T1 (de) | 1990-08-29 | 1997-09-15 | Genpharm Int | Produktion und nützung nicht-menschliche transgentiere zur produktion heterologe antikörper |
| US5877397A (en) | 1990-08-29 | 1999-03-02 | Genpharm International Inc. | Transgenic non-human animals capable of producing heterologous antibodies of various isotypes |
| US5814318A (en) | 1990-08-29 | 1998-09-29 | Genpharm International Inc. | Transgenic non-human animals for producing heterologous antibodies |
| US6300129B1 (en) | 1990-08-29 | 2001-10-09 | Genpharm International | Transgenic non-human animals for producing heterologous antibodies |
| US5874299A (en) | 1990-08-29 | 1999-02-23 | Genpharm International, Inc. | Transgenic non-human animals capable of producing heterologous antibodies |
| US5633425A (en) | 1990-08-29 | 1997-05-27 | Genpharm International, Inc. | Transgenic non-human animals capable of producing heterologous antibodies |
| US5770429A (en) | 1990-08-29 | 1998-06-23 | Genpharm International, Inc. | Transgenic non-human animals capable of producing heterologous antibodies |
| US6255458B1 (en) | 1990-08-29 | 2001-07-03 | Genpharm International | High affinity human antibodies and human antibodies against digoxin |
| US5545806A (en) | 1990-08-29 | 1996-08-13 | Genpharm International, Inc. | Ransgenic non-human animals for producing heterologous antibodies |
| US5661016A (en) | 1990-08-29 | 1997-08-26 | Genpharm International Inc. | Transgenic non-human animals capable of producing heterologous antibodies of various isotypes |
| US5789650A (en) | 1990-08-29 | 1998-08-04 | Genpharm International, Inc. | Transgenic non-human animals for producing heterologous antibodies |
| US5625126A (en) | 1990-08-29 | 1997-04-29 | Genpharm International, Inc. | Transgenic non-human animals for producing heterologous antibodies |
| US5892112A (en) | 1990-11-21 | 1999-04-06 | Glycomed Incorporated | Process for preparing synthetic matrix metalloprotease inhibitors |
| DK0575319T3 (da) | 1991-03-11 | 2000-07-10 | Univ Georgia Res Found | Kloning og ekspression af Renilla-luciferase |
| EP0590076A4 (en) | 1991-06-14 | 1997-02-12 | Dnx Corp | Production of human hemoglobin in transgenic pigs |
| JPH06508880A (ja) | 1991-07-08 | 1994-10-06 | ユニバーシティ オブ マサチューセッツ アット アムハースト | サーモトロピック液晶セグメント化ブロックコポリマー |
| US5262522A (en) | 1991-11-22 | 1993-11-16 | Immunex Corporation | Receptor for oncostatin M and leukemia inhibitory factor |
| US5521184A (en) | 1992-04-03 | 1996-05-28 | Ciba-Geigy Corporation | Pyrimidine derivatives and processes for the preparation thereof |
| ES2301158T3 (es) | 1992-07-24 | 2008-06-16 | Amgen Fremont Inc. | Produccion de anticuerpos xenogenicos. |
| PT672141E (pt) | 1992-10-23 | 2003-09-30 | Immunex Corp | Metodos de preparacao de proteinas oligomericas soluveis |
| JP3560609B2 (ja) | 1992-11-13 | 2004-09-02 | イミュネックス・コーポレーション | Elkリガンドと呼ばれる新規なサイトカイン |
| US5629327A (en) | 1993-03-01 | 1997-05-13 | Childrens Hospital Medical Center Corp. | Methods and compositions for inhibition of angiogenesis |
| US5457035A (en) | 1993-07-23 | 1995-10-10 | Immunex Corporation | Cytokine which is a ligand for OX40 |
| US5516658A (en) | 1993-08-20 | 1996-05-14 | Immunex Corporation | DNA encoding cytokines that bind the cell surface receptor hek |
| WO1995007463A1 (en) | 1993-09-10 | 1995-03-16 | The Trustees Of Columbia University In The City Of New York | Uses of green fluorescent protein |
| US5700823A (en) | 1994-01-07 | 1997-12-23 | Sugen, Inc. | Treatment of platelet derived growth factor related disorders such as cancers |
| IL112248A0 (en) | 1994-01-25 | 1995-03-30 | Warner Lambert Co | Tricyclic heteroaromatic compounds and pharmaceutical compositions containing them |
| WO1995021191A1 (en) | 1994-02-04 | 1995-08-10 | William Ward | Bioluminescent indicator based upon the expression of a gene for a modified green-fluorescent protein |
| EP1464706A3 (en) | 1994-04-15 | 2004-11-03 | Amgen Inc., | HEK5, HEK7, HEK8, HEK11, EPH-like receptor protein tyrosine kinases |
| US6303769B1 (en) | 1994-07-08 | 2001-10-16 | Immunex Corporation | Lerk-5 dna |
| US5919905A (en) | 1994-10-05 | 1999-07-06 | Immunex Corporation | Cytokine designated LERK-6 |
| US5814464A (en) | 1994-10-07 | 1998-09-29 | Regeneron Pharma | Nucleic acids encoding TIE-2 ligand-2 |
| US5777079A (en) | 1994-11-10 | 1998-07-07 | The Regents Of The University Of California | Modified green fluorescent proteins |
| US6057124A (en) | 1995-01-27 | 2000-05-02 | Amgen Inc. | Nucleic acids encoding ligands for HEK4 receptors |
| GB9508538D0 (en) | 1995-04-27 | 1995-06-14 | Zeneca Ltd | Quinazoline derivatives |
| CA2761116A1 (en) | 1995-04-27 | 1996-10-31 | Amgen Fremont Inc. | Human antibodies derived from immunized xenomice |
| US5747498A (en) | 1996-05-28 | 1998-05-05 | Pfizer Inc. | Alkynyl and azido-substituted 4-anilinoquinazolines |
| US5880141A (en) | 1995-06-07 | 1999-03-09 | Sugen, Inc. | Benzylidene-Z-indoline compounds for the treatment of disease |
| DE19534177A1 (de) | 1995-09-15 | 1997-03-20 | Merck Patent Gmbh | Cyclische Adhäsionsinhibitoren |
| US5874304A (en) | 1996-01-18 | 1999-02-23 | University Of Florida Research Foundation, Inc. | Humanized green fluorescent protein genes and methods |
| US5804387A (en) | 1996-02-01 | 1998-09-08 | The Board Of Trustees Of The Leland Stanford Junior University | FACS-optimized mutants of the green fluorescent protein (GFP) |
| US5876995A (en) | 1996-02-06 | 1999-03-02 | Bryan; Bruce | Bioluminescent novelty items |
| TW555765B (en) | 1996-07-09 | 2003-10-01 | Amgen Inc | Low molecular weight soluble tumor necrosis factor type-I and type-II proteins |
| DE69716916T2 (de) | 1996-07-13 | 2003-07-03 | Glaxo Group Ltd | Kondensierte heterozyklische verbindungen als protein kinase inhibitoren |
| US5925558A (en) | 1996-07-16 | 1999-07-20 | The Regents Of The University Of California | Assays for protein kinases using fluorescent protein substrates |
| ID18494A (id) | 1996-10-02 | 1998-04-16 | Novartis Ag | Turunan pirazola leburan dan proses pembuatannya |
| US5976796A (en) | 1996-10-04 | 1999-11-02 | Loma Linda University | Construction and expression of renilla luciferase and green fluorescent protein fusion genes |
| KR20080059467A (ko) | 1996-12-03 | 2008-06-27 | 아브게닉스, 인크. | 복수의 vh 및 vk 부위를 함유하는 사람 면역글로불린유전자좌를 갖는 형질전환된 포유류 및 이로부터 생성된항체 |
| IL129767A0 (en) | 1996-12-12 | 2000-02-29 | Prolume Ltd | Apparatus and method for detecting and identifying infectious agents |
| JP4138013B2 (ja) | 1996-12-23 | 2008-08-20 | イミュネックス・コーポレーション | Tnfスーパーファミリーのメンバーであるnf−kappa bの受容体アクティベーターに対するリガンド |
| CA2196496A1 (en) | 1997-01-31 | 1998-07-31 | Stephen William Watson Michnick | Protein fragment complementation assay for the detection of protein-protein interactions |
| CO4950519A1 (es) | 1997-02-13 | 2000-09-01 | Novartis Ag | Ftalazinas, preparaciones farmaceuticas que las comprenden y proceso para su preparacion |
| US6342220B1 (en) | 1997-08-25 | 2002-01-29 | Genentech, Inc. | Agonist antibodies |
| JP4891477B2 (ja) | 1997-10-02 | 2012-03-07 | マックス−プランク−ゲゼルシャフト ツール フォーデルング デル ヴィッセンシャフテン エー.ヴェー. | 血管新生及び/または既存細動脈網から側枝動脈及び/または他の動脈の発達の調節に関する方法 |
| GB9800569D0 (en) | 1998-01-12 | 1998-03-11 | Glaxo Group Ltd | Heterocyclic compounds |
| WO1999045009A1 (en) | 1998-03-04 | 1999-09-10 | Bristol-Myers Squibb Company | Heterocyclo-substituted imidazopyrazine protein tyrosine kinase inhibitors |
| US6232107B1 (en) | 1998-03-27 | 2001-05-15 | Bruce J. Bryan | Luciferases, fluorescent proteins, nucleic acids encoding the luciferases and fluorescent proteins and the use thereof in diagnostics, high throughput screening and novelty items |
| WO1999061422A1 (en) | 1998-05-29 | 1999-12-02 | Sugen, Inc. | Pyrrole substituted 2-indolinone protein kinase inhibitors |
| UA60365C2 (uk) | 1998-06-04 | 2003-10-15 | Пфайзер Продактс Інк. | Похідні ізотіазолу, спосіб їх одержання, фармацевтична композиція та спосіб лікування гіперпроліферативного захворювання у ссавця |
| EP1097147A4 (en) | 1998-07-10 | 2001-11-21 | Merck & Co Inc | NEW ANGIOGENESIS INHIBITORS |
| CA2341029A1 (en) | 1998-08-17 | 2000-02-24 | Abgenix, Inc. | Generation of modified molecules with increased serum half-lives |
| CA2341409A1 (en) | 1998-08-31 | 2000-03-09 | Merck And Co., Inc. | Novel angiogenesis inhibitors |
| WO2000041698A1 (en) | 1999-01-13 | 2000-07-20 | Bayer Corporation | φ-CARBOXY ARYL SUBSTITUTED DIPHENYL UREAS AS p38 KINASE INHIBITORS |
| CA2366857C (en) | 1999-03-30 | 2010-12-14 | Novartis Ag | Phthalazine derivatives for treating inflammatory diseases |
| US6521424B2 (en) | 1999-06-07 | 2003-02-18 | Immunex Corporation | Recombinant expression of Tek antagonists |
| NZ516258A (en) | 1999-06-07 | 2004-02-27 | Immunex Corp | Tek antagonists |
| AU779892B2 (en) | 1999-08-27 | 2005-02-17 | European Molecular Biology Laboratory | Modified adenoviral fibre and uses thereof |
| DK1223980T3 (da) | 1999-10-28 | 2003-09-15 | Seyedhossein Aharinejad | Anvendelse af CSF-1-inhibitorer |
| DK1676845T3 (da) | 1999-11-05 | 2008-09-15 | Astrazeneca Ab | Nye quinazolinderivater |
| DK1233943T3 (da) | 1999-11-24 | 2011-08-15 | Sugen Inc | Ioniserbare indolinon derivater og anvendelse deraf som PTK ligander |
| US6515004B1 (en) | 1999-12-15 | 2003-02-04 | Bristol-Myers Squibb Company | N-[5-[[[5-alkyl-2-oxazolyl]methyl]thio]-2-thiazolyl]-carboxamide inhibitors of cyclin dependent kinases |
| US6727225B2 (en) | 1999-12-20 | 2004-04-27 | Immunex Corporation | TWEAK receptor |
| AU2001247219B2 (en) | 2000-02-25 | 2007-01-04 | Immunex Corporation | Integrin antagonists |
| US6630500B2 (en) | 2000-08-25 | 2003-10-07 | Cephalon, Inc. | Selected fused pyrrolocarbazoles |
| CZ304059B6 (cs) | 2000-12-21 | 2013-09-11 | Glaxo Group Limited | Deriváty pyrimidinu a farmaceutický prostredek |
| US7102009B2 (en) | 2001-01-12 | 2006-09-05 | Amgen Inc. | Substituted amine derivatives and methods of use |
| US6995162B2 (en) | 2001-01-12 | 2006-02-07 | Amgen Inc. | Substituted alkylamine derivatives and methods of use |
| US20020147198A1 (en) | 2001-01-12 | 2002-10-10 | Guoqing Chen | Substituted arylamine derivatives and methods of use |
| US7105682B2 (en) | 2001-01-12 | 2006-09-12 | Amgen Inc. | Substituted amine derivatives and methods of use |
| US6878714B2 (en) | 2001-01-12 | 2005-04-12 | Amgen Inc. | Substituted alkylamine derivatives and methods of use |
| TR201809008T4 (tr) | 2001-06-26 | 2018-07-23 | Amgen Fremont Inc | Opgl ye karşi antikorlar. |
| US7521053B2 (en) | 2001-10-11 | 2009-04-21 | Amgen Inc. | Angiopoietin-2 specific binding agents |
| US7205275B2 (en) | 2001-10-11 | 2007-04-17 | Amgen Inc. | Methods of treatment using specific binding agents of human angiopoietin-2 |
| US7138370B2 (en) | 2001-10-11 | 2006-11-21 | Amgen Inc. | Specific binding agents of human angiopoietin-2 |
| JP2005530490A (ja) * | 2002-03-29 | 2005-10-13 | シェーリング コーポレイション | インターロイキン−5に対するヒトモノクローナル抗体および方法およびこれを含む組成物 |
| PL375041A1 (en) | 2002-04-05 | 2005-11-14 | Amgen Inc. | Human anti-opgl neutralizing antibodies as selective opgl pathway inhibitors |
| US7307088B2 (en) | 2002-07-09 | 2007-12-11 | Amgen Inc. | Substituted anthranilic amide derivatives and methods of use |
| TWI329112B (en) | 2002-07-19 | 2010-08-21 | Bristol Myers Squibb Co | Novel inhibitors of kinases |
| AR045563A1 (es) * | 2003-09-10 | 2005-11-02 | Warner Lambert Co | Anticuerpos dirigidos a m-csf |
| WO2013180295A1 (ja) | 2012-06-01 | 2013-12-05 | 日本電信電話株式会社 | パケット転送処理方法およびパケット転送処理装置 |
| US9300829B2 (en) | 2014-04-04 | 2016-03-29 | Canon Kabushiki Kaisha | Image reading apparatus and correction method thereof |
-
2008
- 2008-08-19 EP EP17198988.2A patent/EP3330292A1/en not_active Withdrawn
- 2008-08-19 KR KR1020107005166A patent/KR101770429B1/ko active IP Right Grant
- 2008-08-19 DK DK08798203.9T patent/DK2188313T3/en active
- 2008-08-19 WO PCT/US2008/073611 patent/WO2009026303A1/en active Application Filing
- 2008-08-19 NO NO08798203A patent/NO2188313T3/no unknown
- 2008-08-19 LT LTEP08798203.9T patent/LT2188313T/lt unknown
- 2008-08-19 MX MX2010001918A patent/MX2010001918A/es active IP Right Grant
- 2008-08-19 SI SI200831892T patent/SI2188313T1/en unknown
- 2008-08-19 ES ES08798203.9T patent/ES2650224T3/es active Active
- 2008-08-19 TW TW097131630A patent/TWI595005B/zh not_active IP Right Cessation
- 2008-08-19 PL PL08798203T patent/PL2188313T3/pl unknown
- 2008-08-19 AU AU2008288974A patent/AU2008288974B2/en not_active Ceased
- 2008-08-19 RS RS20171242A patent/RS56743B1/sr unknown
- 2008-08-19 EP EP08798203.9A patent/EP2188313B1/en active Active
- 2008-08-19 CA CA2696761A patent/CA2696761C/en active Active
- 2008-08-19 PT PT87982039T patent/PT2188313T/pt unknown
- 2008-08-19 CL CL2008002444A patent/CL2008002444A1/es unknown
- 2008-08-19 EA EA201000357A patent/EA023555B1/ru active IP Right Revival
- 2008-08-19 SG SG10201500328QA patent/SG10201500328QA/en unknown
- 2008-08-19 HU HUE08798203A patent/HUE037265T2/hu unknown
- 2008-08-19 NZ NZ583282A patent/NZ583282A/xx not_active IP Right Cessation
- 2008-08-19 EA EA201591355A patent/EA201591355A1/ru unknown
- 2008-08-19 JP JP2010521980A patent/JP5718640B2/ja active Active
- 2008-08-19 CN CN200880103384.2A patent/CN101802008B/zh active Active
- 2008-08-19 EP EP20120188326 patent/EP2592093A1/en not_active Withdrawn
- 2008-08-19 BR BRPI0815368-0A2A patent/BRPI0815368A2/pt not_active IP Right Cessation
- 2008-08-19 EP EP20120188330 patent/EP2589610A1/en not_active Withdrawn
- 2008-08-20 US US12/195,169 patent/US8182813B2/en active Active
- 2008-08-21 AR ARP080103651 patent/AR068347A1/es not_active Application Discontinuation
- 2008-08-21 PE PE2008001418A patent/PE20091004A1/es not_active Application Discontinuation
-
2010
- 2010-02-18 CR CR11282A patent/CR11282A/es unknown
- 2010-03-03 ZA ZA2010/01541A patent/ZA201001541B/en unknown
-
2012
- 2012-05-08 US US13/466,850 patent/US8513199B2/en active Active
-
2013
- 2013-08-14 US US13/967,037 patent/US9303084B2/en active Active
-
2014
- 2014-12-17 JP JP2014254993A patent/JP6189281B2/ja active Active
-
2016
- 2016-02-19 US US15/048,777 patent/US9988457B2/en active Active
-
2017
- 2017-08-01 JP JP2017148838A patent/JP2018007683A/ja not_active Withdrawn
- 2017-11-13 HR HRP20171741TT patent/HRP20171741T1/hr unknown
- 2017-12-05 CY CY20171101276T patent/CY1119755T1/el unknown
Patent Citations (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2004045532A2 (en) | 2002-11-15 | 2004-06-03 | Chiron Corporation | Methods for preventing and treating cancer metastasis and bone loss associated with cancer metastasis |
Non-Patent Citations (1)
| Title |
|---|
| Blood. Vol. 73, No. 7, pp. 1786-1793 (1989) |
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| KR101770429B1 (ko) | 인간 c-fms 항원 결합 단백질 | |
| KR101359152B1 (ko) | 종양 괴사 인자(tnf)-관련 세포사멸-유도성리간드 수용체 2 폴리펩티드 및 항체 | |
| TWI476206B (zh) | 對肝細胞生長因子具專一性之結合劑 | |
| JP6367233B2 (ja) | 抗pdgfr−ベータ抗体及びそれらの使用 | |
| JP2018150305A (ja) | T細胞、免疫グロブリンドメインおよびムチンドメイン1(tim−1)抗原に対する抗体およびその使用。 | |
| CN113248614A (zh) | 抗pd-1的人抗体 | |
| JP6527508B2 (ja) | αVβ6を過剰発現する癌を治療及び診断する方法 | |
| KR20110031152A (ko) | Il-6에 대한 항체 및 그의 용도 | |
| US20100260769A1 (en) | Endosialin binding molecules | |
| CN101300273A (zh) | Trail受体2多肽和抗体 | |
| KR20240021162A (ko) | Alpha 5 베타 1 인테그린 결합제 및 이의 용도 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A201 | Request for examination | ||
| E902 | Notification of reason for refusal | ||
| E90F | Notification of reason for final refusal | ||
| E701 | Decision to grant or registration of patent right | ||
| GRNT | Written decision to grant |