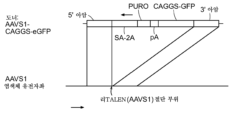
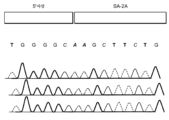
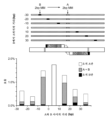
KR20230096134A - 게놈 조작 - Google Patents
게놈 조작 Download PDFInfo
- Publication number
- KR20230096134A KR20230096134A KR1020237020511A KR20237020511A KR20230096134A KR 20230096134 A KR20230096134 A KR 20230096134A KR 1020237020511 A KR1020237020511 A KR 1020237020511A KR 20237020511 A KR20237020511 A KR 20237020511A KR 20230096134 A KR20230096134 A KR 20230096134A
- Authority
- KR
- South Korea
- Prior art keywords
- dna
- artificial
- sequence
- leu
- cells
- Prior art date
Links
- 238000010362 genome editing Methods 0.000 title description 53
- 238000010459 TALEN Methods 0.000 claims abstract description 66
- 238000000034 method Methods 0.000 claims abstract description 43
- 108010043645 Transcription Activator-Like Effector Nucleases Proteins 0.000 claims abstract description 21
- 230000006780 non-homologous end joining Effects 0.000 claims description 56
- 108091033409 CRISPR Proteins 0.000 abstract description 66
- 108091081062 Repeated sequence (DNA) Proteins 0.000 abstract description 10
- 108020004414 DNA Proteins 0.000 description 385
- 102000053602 DNA Human genes 0.000 description 385
- 210000004027 cell Anatomy 0.000 description 185
- 150000007523 nucleic acids Chemical class 0.000 description 97
- 230000008685 targeting Effects 0.000 description 88
- 102000039446 nucleic acids Human genes 0.000 description 84
- 108020004707 nucleic acids Proteins 0.000 description 84
- 230000034431 double-strand break repair via homologous recombination Effects 0.000 description 79
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 58
- 102000004190 Enzymes Human genes 0.000 description 55
- 108090000790 Enzymes Proteins 0.000 description 55
- 229940088598 enzyme Drugs 0.000 description 55
- 108020005004 Guide RNA Proteins 0.000 description 51
- 210000000130 stem cell Anatomy 0.000 description 43
- 101710163270 Nuclease Proteins 0.000 description 36
- 238000006243 chemical reaction Methods 0.000 description 35
- 239000013598 vector Substances 0.000 description 34
- 102000052510 DNA-Binding Proteins Human genes 0.000 description 31
- 108010073062 Transcription Activator-Like Effectors Proteins 0.000 description 28
- 239000002609 medium Substances 0.000 description 28
- 238000003780 insertion Methods 0.000 description 27
- 238000003776 cleavage reaction Methods 0.000 description 26
- 230000037431 insertion Effects 0.000 description 26
- 230000007017 scission Effects 0.000 description 26
- 230000000694 effects Effects 0.000 description 23
- 108090000623 proteins and genes Proteins 0.000 description 23
- 238000001890 transfection Methods 0.000 description 23
- 230000000295 complement effect Effects 0.000 description 21
- 102100032912 CD44 antigen Human genes 0.000 description 19
- 101000868273 Homo sapiens CD44 antigen Proteins 0.000 description 19
- 230000001404 mediated effect Effects 0.000 description 19
- 239000013612 plasmid Substances 0.000 description 19
- 230000008045 co-localization Effects 0.000 description 18
- 239000000203 mixture Substances 0.000 description 18
- 101710096438 DNA-binding protein Proteins 0.000 description 17
- 108091028043 Nucleic acid sequence Proteins 0.000 description 17
- 238000013461 design Methods 0.000 description 15
- 239000000539 dimer Substances 0.000 description 15
- 230000006801 homologous recombination Effects 0.000 description 15
- 238000002744 homologous recombination Methods 0.000 description 15
- 108700020911 DNA-Binding Proteins Proteins 0.000 description 14
- 230000035772 mutation Effects 0.000 description 14
- 102100035875 C-C chemokine receptor type 5 Human genes 0.000 description 13
- 101710149870 C-C chemokine receptor type 5 Proteins 0.000 description 13
- 230000005782 double-strand break Effects 0.000 description 13
- 210000004263 induced pluripotent stem cell Anatomy 0.000 description 13
- 238000012217 deletion Methods 0.000 description 12
- 230000037430 deletion Effects 0.000 description 12
- 238000009826 distribution Methods 0.000 description 12
- RXWNCPJZOCPEPQ-NVWDDTSBSA-N puromycin Chemical compound C1=CC(OC)=CC=C1C[C@H](N)C(=O)N[C@H]1[C@@H](O)[C@H](N2C3=NC=NC(=C3N=C2)N(C)C)O[C@@H]1CO RXWNCPJZOCPEPQ-NVWDDTSBSA-N 0.000 description 12
- 102000004533 Endonucleases Human genes 0.000 description 11
- 108010042407 Endonucleases Proteins 0.000 description 11
- 108060002716 Exonuclease Proteins 0.000 description 11
- 102000013165 exonuclease Human genes 0.000 description 11
- 102000040650 (ribonucleotides)n+m Human genes 0.000 description 10
- 101001000998 Homo sapiens Protein phosphatase 1 regulatory subunit 12C Proteins 0.000 description 10
- 102100035620 Protein phosphatase 1 regulatory subunit 12C Human genes 0.000 description 10
- 238000012360 testing method Methods 0.000 description 10
- 238000010354 CRISPR gene editing Methods 0.000 description 9
- 230000004568 DNA-binding Effects 0.000 description 9
- 108010008532 Deoxyribonuclease I Proteins 0.000 description 9
- 102000007260 Deoxyribonuclease I Human genes 0.000 description 9
- 108091034117 Oligonucleotide Proteins 0.000 description 9
- 238000002474 experimental method Methods 0.000 description 9
- 239000002773 nucleotide Substances 0.000 description 9
- 125000003729 nucleotide group Chemical group 0.000 description 9
- 239000000047 product Substances 0.000 description 9
- 230000008439 repair process Effects 0.000 description 9
- 108090000608 Phosphoric Monoester Hydrolases Proteins 0.000 description 8
- 102000004160 Phosphoric Monoester Hydrolases Human genes 0.000 description 8
- 238000003556 assay Methods 0.000 description 8
- 238000001943 fluorescence-activated cell sorting Methods 0.000 description 8
- 230000035945 sensitivity Effects 0.000 description 8
- 150000001413 amino acids Chemical group 0.000 description 7
- 230000027455 binding Effects 0.000 description 7
- 239000006285 cell suspension Substances 0.000 description 7
- 238000011156 evaluation Methods 0.000 description 7
- 108010082117 matrigel Proteins 0.000 description 7
- 230000004048 modification Effects 0.000 description 7
- 238000012986 modification Methods 0.000 description 7
- 238000011160 research Methods 0.000 description 7
- 238000012163 sequencing technique Methods 0.000 description 7
- 238000012546 transfer Methods 0.000 description 7
- OZFAFGSSMRRTDW-UHFFFAOYSA-N (2,4-dichlorophenyl) benzenesulfonate Chemical compound ClC1=CC(Cl)=CC=C1OS(=O)(=O)C1=CC=CC=C1 OZFAFGSSMRRTDW-UHFFFAOYSA-N 0.000 description 6
- 108010014303 DNA-directed DNA polymerase Proteins 0.000 description 6
- 102000016928 DNA-directed DNA polymerase Human genes 0.000 description 6
- 239000012591 Dulbecco’s Phosphate Buffered Saline Substances 0.000 description 6
- 206010043276 Teratoma Diseases 0.000 description 6
- 102000008579 Transposases Human genes 0.000 description 6
- 108010020764 Transposases Proteins 0.000 description 6
- KOSRFJWDECSPRO-UHFFFAOYSA-N alpha-L-glutamyl-L-glutamic acid Natural products OC(=O)CCC(N)C(=O)NC(CCC(O)=O)C(O)=O KOSRFJWDECSPRO-UHFFFAOYSA-N 0.000 description 6
- 238000001514 detection method Methods 0.000 description 6
- 239000012634 fragment Substances 0.000 description 6
- 238000010348 incorporation Methods 0.000 description 6
- 239000003112 inhibitor Substances 0.000 description 6
- 108010034529 leucyl-lysine Proteins 0.000 description 6
- 239000000178 monomer Substances 0.000 description 6
- 238000007481 next generation sequencing Methods 0.000 description 6
- 102000004169 proteins and genes Human genes 0.000 description 6
- 229950010131 puromycin Drugs 0.000 description 6
- 239000011435 rock Substances 0.000 description 6
- 102000012410 DNA Ligases Human genes 0.000 description 5
- 108010061982 DNA Ligases Proteins 0.000 description 5
- 108091029865 Exogenous DNA Proteins 0.000 description 5
- 101000825726 Homo sapiens Structural maintenance of chromosomes protein 4 Proteins 0.000 description 5
- 241000713666 Lentivirus Species 0.000 description 5
- 241000193996 Streptococcus pyogenes Species 0.000 description 5
- 102100022842 Structural maintenance of chromosomes protein 4 Human genes 0.000 description 5
- 230000004075 alteration Effects 0.000 description 5
- 238000004458 analytical method Methods 0.000 description 5
- 108010092854 aspartyllysine Proteins 0.000 description 5
- 210000004413 cardiac myocyte Anatomy 0.000 description 5
- 238000010367 cloning Methods 0.000 description 5
- 230000001973 epigenetic effect Effects 0.000 description 5
- 108010055341 glutamyl-glutamic acid Proteins 0.000 description 5
- 238000011534 incubation Methods 0.000 description 5
- 230000001939 inductive effect Effects 0.000 description 5
- 108020004999 messenger RNA Proteins 0.000 description 5
- 238000012545 processing Methods 0.000 description 5
- 230000003252 repetitive effect Effects 0.000 description 5
- 230000002441 reversible effect Effects 0.000 description 5
- 239000000243 solution Substances 0.000 description 5
- 238000011144 upstream manufacturing Methods 0.000 description 5
- 108700028369 Alleles Proteins 0.000 description 4
- 108091026890 Coding region Proteins 0.000 description 4
- 230000007018 DNA scission Effects 0.000 description 4
- 239000006144 Dulbecco’s modified Eagle's medium Substances 0.000 description 4
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 4
- 241000589602 Francisella tularensis Species 0.000 description 4
- XVZCXCTYGHPNEM-UHFFFAOYSA-N Leu-Leu-Pro Natural products CC(C)CC(N)C(=O)NC(CC(C)C)C(=O)N1CCCC1C(O)=O XVZCXCTYGHPNEM-UHFFFAOYSA-N 0.000 description 4
- 108090000364 Ligases Proteins 0.000 description 4
- 102000003960 Ligases Human genes 0.000 description 4
- 241000194017 Streptococcus Species 0.000 description 4
- 241000700605 Viruses Species 0.000 description 4
- 210000004102 animal cell Anatomy 0.000 description 4
- 238000013459 approach Methods 0.000 description 4
- 230000010310 bacterial transformation Effects 0.000 description 4
- 239000000872 buffer Substances 0.000 description 4
- 230000008859 change Effects 0.000 description 4
- 230000001351 cycling effect Effects 0.000 description 4
- 239000012091 fetal bovine serum Substances 0.000 description 4
- 238000000684 flow cytometry Methods 0.000 description 4
- 229940118764 francisella tularensis Drugs 0.000 description 4
- 230000004927 fusion Effects 0.000 description 4
- 230000002068 genetic effect Effects 0.000 description 4
- 108010050848 glycylleucine Proteins 0.000 description 4
- 210000005260 human cell Anatomy 0.000 description 4
- 238000000746 purification Methods 0.000 description 4
- 230000000392 somatic effect Effects 0.000 description 4
- UCSJYZPVAKXKNQ-HZYVHMACSA-N streptomycin Chemical compound CN[C@H]1[C@H](O)[C@@H](O)[C@H](CO)O[C@H]1O[C@@H]1[C@](C=O)(O)[C@H](C)O[C@H]1O[C@@H]1[C@@H](NC(N)=N)[C@H](O)[C@@H](NC(N)=N)[C@H](O)[C@H]1O UCSJYZPVAKXKNQ-HZYVHMACSA-N 0.000 description 4
- 238000010361 transduction Methods 0.000 description 4
- 230000026683 transduction Effects 0.000 description 4
- 108091093088 Amplicon Proteins 0.000 description 3
- 108010063905 Ampligase Proteins 0.000 description 3
- 108091079001 CRISPR RNA Proteins 0.000 description 3
- 241000196324 Embryophyta Species 0.000 description 3
- JBCLFWXMTIKCCB-UHFFFAOYSA-N H-Gly-Phe-OH Natural products NCC(=O)NC(C(O)=O)CC1=CC=CC=C1 JBCLFWXMTIKCCB-UHFFFAOYSA-N 0.000 description 3
- IRMLZWSRWSGTOP-CIUDSAMLSA-N Leu-Ser-Ala Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CO)C(=O)N[C@@H](C)C(O)=O IRMLZWSRWSGTOP-CIUDSAMLSA-N 0.000 description 3
- DRRXXZBXDMLGFC-IHRRRGAJSA-N Lys-Val-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](C(C)C)NC(=O)[C@@H](N)CCCCN DRRXXZBXDMLGFC-IHRRRGAJSA-N 0.000 description 3
- 201000009906 Meningitis Diseases 0.000 description 3
- 241000699666 Mus <mouse, genus> Species 0.000 description 3
- 241000588653 Neisseria Species 0.000 description 3
- 241000700159 Rattus Species 0.000 description 3
- LTFLDDDGWOVIHY-NAKRPEOUSA-N Val-Ala-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H](C)NC(=O)[C@H](C(C)C)N LTFLDDDGWOVIHY-NAKRPEOUSA-N 0.000 description 3
- 230000003213 activating effect Effects 0.000 description 3
- 230000003044 adaptive effect Effects 0.000 description 3
- 108010062796 arginyllysine Proteins 0.000 description 3
- 230000000712 assembly Effects 0.000 description 3
- 238000000429 assembly Methods 0.000 description 3
- 230000007423 decrease Effects 0.000 description 3
- 230000003247 decreasing effect Effects 0.000 description 3
- 108010007093 dispase Proteins 0.000 description 3
- 230000006870 function Effects 0.000 description 3
- 239000000499 gel Substances 0.000 description 3
- 239000001963 growth medium Substances 0.000 description 3
- 108010025306 histidylleucine Proteins 0.000 description 3
- 238000000338 in vitro Methods 0.000 description 3
- 108010057821 leucylproline Proteins 0.000 description 3
- 239000000463 material Substances 0.000 description 3
- 238000002360 preparation method Methods 0.000 description 3
- 230000008569 process Effects 0.000 description 3
- 108090000765 processed proteins & peptides Proteins 0.000 description 3
- 230000008263 repair mechanism Effects 0.000 description 3
- 108091008146 restriction endonucleases Proteins 0.000 description 3
- 238000012552 review Methods 0.000 description 3
- 238000007480 sanger sequencing Methods 0.000 description 3
- 238000012216 screening Methods 0.000 description 3
- 210000001082 somatic cell Anatomy 0.000 description 3
- 125000006850 spacer group Chemical group 0.000 description 3
- 241000894007 species Species 0.000 description 3
- 239000006228 supernatant Substances 0.000 description 3
- 210000001519 tissue Anatomy 0.000 description 3
- 230000002103 transcriptional effect Effects 0.000 description 3
- 230000009466 transformation Effects 0.000 description 3
- 239000003981 vehicle Substances 0.000 description 3
- 230000003612 virological effect Effects 0.000 description 3
- XVZCXCTYGHPNEM-IHRRRGAJSA-N (2s)-1-[(2s)-2-[[(2s)-2-amino-4-methylpentanoyl]amino]-4-methylpentanoyl]pyrrolidine-2-carboxylic acid Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N1CCC[C@H]1C(O)=O XVZCXCTYGHPNEM-IHRRRGAJSA-N 0.000 description 2
- GUAHPAJOXVYFON-ZETCQYMHSA-N (8S)-8-amino-7-oxononanoic acid zwitterion Chemical compound C[C@H](N)C(=O)CCCCCC(O)=O GUAHPAJOXVYFON-ZETCQYMHSA-N 0.000 description 2
- 241000702462 Akkermansia muciniphila Species 0.000 description 2
- VWEWCZSUWOEEFM-WDSKDSINSA-N Ala-Gly-Ala-Gly Chemical compound C[C@H](N)C(=O)NCC(=O)N[C@@H](C)C(=O)NCC(O)=O VWEWCZSUWOEEFM-WDSKDSINSA-N 0.000 description 2
- MDNAVFBZPROEHO-UHFFFAOYSA-N Ala-Lys-Val Natural products CC(C)C(C(O)=O)NC(=O)C(NC(=O)C(C)N)CCCCN MDNAVFBZPROEHO-UHFFFAOYSA-N 0.000 description 2
- VNFWDYWTSHFRRG-SRVKXCTJSA-N Arg-Gln-Leu Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(C)C)C(O)=O VNFWDYWTSHFRRG-SRVKXCTJSA-N 0.000 description 2
- OTZMRMHZCMZOJZ-SRVKXCTJSA-N Arg-Leu-Glu Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(O)=O)C(O)=O OTZMRMHZCMZOJZ-SRVKXCTJSA-N 0.000 description 2
- IZSMEUDYADKZTJ-KJEVXHAQSA-N Arg-Tyr-Thr Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H]([C@@H](C)O)C(O)=O IZSMEUDYADKZTJ-KJEVXHAQSA-N 0.000 description 2
- BDMIFVIWCNLDCT-CIUDSAMLSA-N Asn-Arg-Glu Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(O)=O)C(O)=O BDMIFVIWCNLDCT-CIUDSAMLSA-N 0.000 description 2
- OPEPUCYIGFEGSW-WDSKDSINSA-N Asn-Gly-Glu Chemical compound [H]N[C@@H](CC(N)=O)C(=O)NCC(=O)N[C@@H](CCC(O)=O)C(O)=O OPEPUCYIGFEGSW-WDSKDSINSA-N 0.000 description 2
- NJSNXIOKBHPFMB-GMOBBJLQSA-N Asn-Pro-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@@H]1CCCN1C(=O)[C@H](CC(=O)N)N NJSNXIOKBHPFMB-GMOBBJLQSA-N 0.000 description 2
- UJGRZQYSNYTCAX-SRVKXCTJSA-N Asp-Leu-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@@H](N)CC(O)=O UJGRZQYSNYTCAX-SRVKXCTJSA-N 0.000 description 2
- LTCKTLYKRMCFOC-KKUMJFAQSA-N Asp-Phe-Leu Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CC(C)C)C(O)=O LTCKTLYKRMCFOC-KKUMJFAQSA-N 0.000 description 2
- 241000894006 Bacteria Species 0.000 description 2
- 108091032955 Bacterial small RNA Proteins 0.000 description 2
- 108091003079 Bovine Serum Albumin Proteins 0.000 description 2
- 241000589875 Campylobacter jejuni Species 0.000 description 2
- 208000031229 Cardiomyopathies Diseases 0.000 description 2
- 241000193155 Clostridium botulinum Species 0.000 description 2
- 108020004705 Codon Proteins 0.000 description 2
- 241000186226 Corynebacterium glutamicum Species 0.000 description 2
- 238000007400 DNA extraction Methods 0.000 description 2
- 230000008836 DNA modification Effects 0.000 description 2
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 2
- 241001338691 Elusimicrobium minutum Species 0.000 description 2
- 241000382842 Flavobacterium psychrophilum Species 0.000 description 2
- WSFSSNUMVMOOMR-UHFFFAOYSA-N Formaldehyde Chemical compound O=C WSFSSNUMVMOOMR-UHFFFAOYSA-N 0.000 description 2
- 241000589601 Francisella Species 0.000 description 2
- UWZLBXOBVKRUFE-HGNGGELXSA-N Gln-Ala-His Chemical compound C[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)NC(=O)[C@H](CCC(=O)N)N UWZLBXOBVKRUFE-HGNGGELXSA-N 0.000 description 2
- PRBLYKYHAJEABA-SRVKXCTJSA-N Gln-Arg-Leu Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(C)C)C(O)=O PRBLYKYHAJEABA-SRVKXCTJSA-N 0.000 description 2
- HTTSBEBKVNEDFE-AUTRQRHGSA-N Glu-Gln-Val Chemical compound CC(C)[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CCC(=O)O)N HTTSBEBKVNEDFE-AUTRQRHGSA-N 0.000 description 2
- MWMJCGBSIORNCD-AVGNSLFASA-N Glu-Leu-Leu Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC(C)C)C(O)=O MWMJCGBSIORNCD-AVGNSLFASA-N 0.000 description 2
- DLISPGXMKZTWQG-IFFSRLJSSA-N Glu-Thr-Val Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C(C)C)C(O)=O DLISPGXMKZTWQG-IFFSRLJSSA-N 0.000 description 2
- LHYJCVCQPWRMKZ-WEDXCCLWSA-N Gly-Leu-Thr Chemical compound [H]NCC(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O LHYJCVCQPWRMKZ-WEDXCCLWSA-N 0.000 description 2
- NTBOEZICHOSJEE-YUMQZZPRSA-N Gly-Lys-Ser Chemical compound [H]NCC(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CO)C(O)=O NTBOEZICHOSJEE-YUMQZZPRSA-N 0.000 description 2
- BCISUQVFDGYZBO-QSFUFRPTSA-N Ile-Val-Asp Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@H](C(O)=O)CC(O)=O BCISUQVFDGYZBO-QSFUFRPTSA-N 0.000 description 2
- KFZMGEQAYNKOFK-UHFFFAOYSA-N Isopropanol Chemical compound CC(C)O KFZMGEQAYNKOFK-UHFFFAOYSA-N 0.000 description 2
- 241000880493 Leptailurus serval Species 0.000 description 2
- WUFYAPWIHCUMLL-CIUDSAMLSA-N Leu-Asn-Ala Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](C)C(O)=O WUFYAPWIHCUMLL-CIUDSAMLSA-N 0.000 description 2
- QKIBIXAQKAFZGL-GUBZILKMSA-N Leu-Cys-Gln Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CS)C(=O)N[C@@H](CCC(N)=O)C(O)=O QKIBIXAQKAFZGL-GUBZILKMSA-N 0.000 description 2
- YRRCOJOXAJNSAX-IHRRRGAJSA-N Leu-Pro-Lys Chemical compound CC(C)C[C@@H](C(=O)N1CCC[C@H]1C(=O)N[C@@H](CCCCN)C(=O)O)N YRRCOJOXAJNSAX-IHRRRGAJSA-N 0.000 description 2
- JDBQSGMJBMPNFT-AVGNSLFASA-N Leu-Pro-Val Chemical compound CC(C)C[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](C(C)C)C(O)=O JDBQSGMJBMPNFT-AVGNSLFASA-N 0.000 description 2
- FACUGMGEFUEBTI-SRVKXCTJSA-N Lys-Asn-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@@H](N)CCCCN FACUGMGEFUEBTI-SRVKXCTJSA-N 0.000 description 2
- DFXQCCBKGUNYGG-GUBZILKMSA-N Lys-Gln-Ala Chemical compound OC(=O)[C@H](C)NC(=O)[C@H](CCC(N)=O)NC(=O)[C@@H](N)CCCCN DFXQCCBKGUNYGG-GUBZILKMSA-N 0.000 description 2
- WVJNGSFKBKOKRV-AJNGGQMLSA-N Lys-Leu-Ile Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O WVJNGSFKBKOKRV-AJNGGQMLSA-N 0.000 description 2
- LJADEBULDNKJNK-IHRRRGAJSA-N Lys-Leu-Val Chemical compound CC(C)C[C@H](NC(=O)[C@@H](N)CCCCN)C(=O)N[C@@H](C(C)C)C(O)=O LJADEBULDNKJNK-IHRRRGAJSA-N 0.000 description 2
- JMNRXRPBHFGXQX-GUBZILKMSA-N Lys-Ser-Glu Chemical compound NCCCC[C@H](N)C(=O)N[C@@H](CO)C(=O)N[C@H](C(O)=O)CCC(O)=O JMNRXRPBHFGXQX-GUBZILKMSA-N 0.000 description 2
- TWRXJAOTZQYOKJ-UHFFFAOYSA-L Magnesium chloride Chemical compound [Mg+2].[Cl-].[Cl-] TWRXJAOTZQYOKJ-UHFFFAOYSA-L 0.000 description 2
- RRIHXWPHQSXHAQ-XUXIUFHCSA-N Met-Ile-Lys Chemical compound CSCC[C@H](N)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CCCCN)C(O)=O RRIHXWPHQSXHAQ-XUXIUFHCSA-N 0.000 description 2
- 108020005196 Mitochondrial DNA Proteins 0.000 description 2
- 241001135743 Mycoplasma penetrans Species 0.000 description 2
- YBAFDPFAUTYYRW-UHFFFAOYSA-N N-L-alpha-glutamyl-L-leucine Natural products CC(C)CC(C(O)=O)NC(=O)C(N)CCC(O)=O YBAFDPFAUTYYRW-UHFFFAOYSA-N 0.000 description 2
- SITLTJHOQZFJGG-UHFFFAOYSA-N N-L-alpha-glutamyl-L-valine Natural products CC(C)C(C(O)=O)NC(=O)C(N)CCC(O)=O SITLTJHOQZFJGG-UHFFFAOYSA-N 0.000 description 2
- 229930182555 Penicillin Natural products 0.000 description 2
- JGSARLDLIJGVTE-MBNYWOFBSA-N Penicillin G Chemical compound N([C@H]1[C@H]2SC([C@@H](N2C1=O)C(O)=O)(C)C)C(=O)CC1=CC=CC=C1 JGSARLDLIJGVTE-MBNYWOFBSA-N 0.000 description 2
- 108091005804 Peptidases Proteins 0.000 description 2
- WEMYTDDMDBLPMI-DKIMLUQUSA-N Phe-Ile-Lys Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CC1=CC=CC=C1)N WEMYTDDMDBLPMI-DKIMLUQUSA-N 0.000 description 2
- IPFXYNKCXYGSSV-KKUMJFAQSA-N Phe-Ser-Lys Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CCCCN)C(=O)O)N IPFXYNKCXYGSSV-KKUMJFAQSA-N 0.000 description 2
- 102100037486 Reverse transcriptase/ribonuclease H Human genes 0.000 description 2
- 241000190950 Rhodopseudomonas palustris Species 0.000 description 2
- MIJWOJAXARLEHA-WDSKDSINSA-N Ser-Gly-Glu Chemical compound OC[C@H](N)C(=O)NCC(=O)N[C@H](C(O)=O)CCC(O)=O MIJWOJAXARLEHA-WDSKDSINSA-N 0.000 description 2
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 2
- 241000194020 Streptococcus thermophilus Species 0.000 description 2
- 241000159619 Tolumonas auensis Species 0.000 description 2
- GULIUBBXCYPDJU-CQDKDKBSSA-N Tyr-Leu-Ala Chemical compound [O-]C(=O)[C@H](C)NC(=O)[C@H](CC(C)C)NC(=O)[C@@H]([NH3+])CC1=CC=C(O)C=C1 GULIUBBXCYPDJU-CQDKDKBSSA-N 0.000 description 2
- BMOFUVHDBROBSE-DCAQKATOSA-N Val-Leu-Cys Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CS)C(=O)O)NC(=O)[C@H](C(C)C)N BMOFUVHDBROBSE-DCAQKATOSA-N 0.000 description 2
- 108020005202 Viral DNA Proteins 0.000 description 2
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 description 2
- 230000004913 activation Effects 0.000 description 2
- 230000001464 adherent effect Effects 0.000 description 2
- 238000000137 annealing Methods 0.000 description 2
- 230000008970 bacterial immunity Effects 0.000 description 2
- 230000008901 benefit Effects 0.000 description 2
- 230000015572 biosynthetic process Effects 0.000 description 2
- 238000004364 calculation method Methods 0.000 description 2
- 239000007795 chemical reaction product Substances 0.000 description 2
- 238000013373 clone screening Methods 0.000 description 2
- 238000010276 construction Methods 0.000 description 2
- 238000012350 deep sequencing Methods 0.000 description 2
- FSXRLASFHBWESK-UHFFFAOYSA-N dipeptide phenylalanyl-tyrosine Natural products C=1C=C(O)C=CC=1CC(C(O)=O)NC(=O)C(N)CC1=CC=CC=C1 FSXRLASFHBWESK-UHFFFAOYSA-N 0.000 description 2
- 239000012636 effector Substances 0.000 description 2
- 238000004520 electroporation Methods 0.000 description 2
- 210000003527 eukaryotic cell Anatomy 0.000 description 2
- 238000013401 experimental design Methods 0.000 description 2
- 108010063718 gamma-glutamylaspartic acid Proteins 0.000 description 2
- 238000001476 gene delivery Methods 0.000 description 2
- 238000013412 genome amplification Methods 0.000 description 2
- 238000003205 genotyping method Methods 0.000 description 2
- 108010015792 glycyllysine Proteins 0.000 description 2
- PCHJSUWPFVWCPO-UHFFFAOYSA-N gold Chemical compound [Au] PCHJSUWPFVWCPO-UHFFFAOYSA-N 0.000 description 2
- 229910052737 gold Inorganic materials 0.000 description 2
- 239000010931 gold Substances 0.000 description 2
- 238000007490 hematoxylin and eosin (H&E) staining Methods 0.000 description 2
- 108010018006 histidylserine Proteins 0.000 description 2
- 230000028993 immune response Effects 0.000 description 2
- 238000012744 immunostaining Methods 0.000 description 2
- 238000011065 in-situ storage Methods 0.000 description 2
- 230000006698 induction Effects 0.000 description 2
- 238000012966 insertion method Methods 0.000 description 2
- 238000011813 knockout mouse model Methods 0.000 description 2
- 108010064235 lysylglycine Proteins 0.000 description 2
- 108010054155 lysyllysine Proteins 0.000 description 2
- 108010017391 lysylvaline Proteins 0.000 description 2
- 239000003550 marker Substances 0.000 description 2
- 230000007246 mechanism Effects 0.000 description 2
- 230000032965 negative regulation of cell volume Effects 0.000 description 2
- 238000005457 optimization Methods 0.000 description 2
- 229940049954 penicillin Drugs 0.000 description 2
- 125000002467 phosphate group Chemical group [H]OP(=O)(O[H])O[*] 0.000 description 2
- BASFCYQUMIYNBI-UHFFFAOYSA-N platinum Chemical compound [Pt] BASFCYQUMIYNBI-UHFFFAOYSA-N 0.000 description 2
- 229920001184 polypeptide Polymers 0.000 description 2
- 102000004196 processed proteins & peptides Human genes 0.000 description 2
- 239000011535 reaction buffer Substances 0.000 description 2
- 230000006798 recombination Effects 0.000 description 2
- 238000005215 recombination Methods 0.000 description 2
- 238000012772 sequence design Methods 0.000 description 2
- 229960005322 streptomycin Drugs 0.000 description 2
- 239000013589 supplement Substances 0.000 description 2
- 238000012353 t test Methods 0.000 description 2
- 108091006106 transcriptional activators Proteins 0.000 description 2
- 238000003151 transfection method Methods 0.000 description 2
- 108010073969 valyllysine Proteins 0.000 description 2
- 230000035899 viability Effects 0.000 description 2
- 210000005253 yeast cell Anatomy 0.000 description 2
- IDGZVZJLYFTXSL-UHFFFAOYSA-N 2-[[2-[(2-amino-4-methylpentanoyl)amino]-3-hydroxypropanoyl]amino]-5-(diaminomethylideneamino)pentanoic acid Chemical compound CC(C)CC(N)C(=O)NC(CO)C(=O)NC(C(O)=O)CCCN=C(N)N IDGZVZJLYFTXSL-UHFFFAOYSA-N 0.000 description 1
- QKNYBSVHEMOAJP-UHFFFAOYSA-N 2-amino-2-(hydroxymethyl)propane-1,3-diol;hydron;chloride Chemical compound Cl.OCC(N)(CO)CO QKNYBSVHEMOAJP-UHFFFAOYSA-N 0.000 description 1
- IDDDVXIUIXWAGJ-DDSAHXNVSA-N 4-[(1r)-1-aminoethyl]-n-pyridin-4-ylcyclohexane-1-carboxamide;dihydrochloride Chemical compound Cl.Cl.C1CC([C@H](N)C)CCC1C(=O)NC1=CC=NC=C1 IDDDVXIUIXWAGJ-DDSAHXNVSA-N 0.000 description 1
- 230000002407 ATP formation Effects 0.000 description 1
- 108091006112 ATPases Proteins 0.000 description 1
- 241001041760 Acidothermus cellulolyticus 11B Species 0.000 description 1
- 241000417230 Actinobacillus succinogenes 130Z Species 0.000 description 1
- 102000057290 Adenosine Triphosphatases Human genes 0.000 description 1
- FJVAQLJNTSUQPY-CIUDSAMLSA-N Ala-Ala-Lys Chemical compound C[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@H](C(O)=O)CCCCN FJVAQLJNTSUQPY-CIUDSAMLSA-N 0.000 description 1
- LWUWMHIOBPTZBA-DCAQKATOSA-N Ala-Arg-Lys Chemical compound NC(=N)NCCC[C@H](NC(=O)[C@@H](N)C)C(=O)N[C@@H](CCCCN)C(O)=O LWUWMHIOBPTZBA-DCAQKATOSA-N 0.000 description 1
- XEXJJJRVTFGWIC-FXQIFTODSA-N Ala-Asn-Arg Chemical compound C[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H](CCCN=C(N)N)C(=O)O)N XEXJJJRVTFGWIC-FXQIFTODSA-N 0.000 description 1
- NXSFUECZFORGOG-CIUDSAMLSA-N Ala-Asn-Leu Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CC(C)C)C(O)=O NXSFUECZFORGOG-CIUDSAMLSA-N 0.000 description 1
- FVSOUJZKYWEFOB-KBIXCLLPSA-N Ala-Gln-Ile Chemical compound CC[C@H](C)[C@@H](C(O)=O)NC(=O)[C@H](CCC(N)=O)NC(=O)[C@H](C)N FVSOUJZKYWEFOB-KBIXCLLPSA-N 0.000 description 1
- YIGLXQRFQVWFEY-NRPADANISA-N Ala-Gln-Val Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](C(C)C)C(O)=O YIGLXQRFQVWFEY-NRPADANISA-N 0.000 description 1
- NWVVKQZOVSTDBQ-CIUDSAMLSA-N Ala-Glu-Arg Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O NWVVKQZOVSTDBQ-CIUDSAMLSA-N 0.000 description 1
- NJPMYXWVWQWCSR-ACZMJKKPSA-N Ala-Glu-Asn Chemical compound C[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(N)=O)C(O)=O NJPMYXWVWQWCSR-ACZMJKKPSA-N 0.000 description 1
- KXEVYGKATAMXJJ-ACZMJKKPSA-N Ala-Glu-Asp Chemical compound C[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(O)=O)C(O)=O KXEVYGKATAMXJJ-ACZMJKKPSA-N 0.000 description 1
- OKEWAFFWMHBGPT-XPUUQOCRSA-N Ala-His-Gly Chemical compound OC(=O)CNC(=O)[C@@H](NC(=O)[C@@H](N)C)CC1=CN=CN1 OKEWAFFWMHBGPT-XPUUQOCRSA-N 0.000 description 1
- PMQXMXAASGFUDX-SRVKXCTJSA-N Ala-Lys-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)[C@H](C)N)CCCCN PMQXMXAASGFUDX-SRVKXCTJSA-N 0.000 description 1
- ZBLQIYPCUWZSRZ-QEJZJMRPSA-N Ala-Phe-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)[C@H](C)N)CC1=CC=CC=C1 ZBLQIYPCUWZSRZ-QEJZJMRPSA-N 0.000 description 1
- KLALXKYLOMZDQT-ZLUOBGJFSA-N Ala-Ser-Asn Chemical compound C[C@H](N)C(=O)N[C@@H](CO)C(=O)N[C@H](C(O)=O)CC(N)=O KLALXKYLOMZDQT-ZLUOBGJFSA-N 0.000 description 1
- NHWYNIZWLJYZAG-XVYDVKMFSA-N Ala-Ser-His Chemical compound C[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)N NHWYNIZWLJYZAG-XVYDVKMFSA-N 0.000 description 1
- HOVPGJUNRLMIOZ-CIUDSAMLSA-N Ala-Ser-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@H](C)N HOVPGJUNRLMIOZ-CIUDSAMLSA-N 0.000 description 1
- VRTOMXFZHGWHIJ-KZVJFYERSA-N Ala-Thr-Arg Chemical compound [H]N[C@@H](C)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O VRTOMXFZHGWHIJ-KZVJFYERSA-N 0.000 description 1
- IOFVWPYSRSCWHI-JXUBOQSCSA-N Ala-Thr-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H]([C@@H](C)O)NC(=O)[C@H](C)N IOFVWPYSRSCWHI-JXUBOQSCSA-N 0.000 description 1
- IETUUAHKCHOQHP-KZVJFYERSA-N Ala-Thr-Val Chemical compound CC(C)[C@H](NC(=O)[C@@H](NC(=O)[C@H](C)N)[C@@H](C)O)C(O)=O IETUUAHKCHOQHP-KZVJFYERSA-N 0.000 description 1
- QRIYOHQJRDHFKF-UWJYBYFXSA-N Ala-Tyr-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)[C@@H](N)C)CC1=CC=C(O)C=C1 QRIYOHQJRDHFKF-UWJYBYFXSA-N 0.000 description 1
- 241000203069 Archaea Species 0.000 description 1
- MUXONAMCEUBVGA-DCAQKATOSA-N Arg-Arg-Gln Chemical compound NC(N)=NCCC[C@H](N)C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CCC(N)=O)C(O)=O MUXONAMCEUBVGA-DCAQKATOSA-N 0.000 description 1
- HJVGMOYJDDXLMI-AVGNSLFASA-N Arg-Arg-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@@H](N)CCCNC(N)=N HJVGMOYJDDXLMI-AVGNSLFASA-N 0.000 description 1
- IIABBYGHLYWVOS-FXQIFTODSA-N Arg-Asn-Ser Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CO)C(O)=O IIABBYGHLYWVOS-FXQIFTODSA-N 0.000 description 1
- RRGPUNYIPJXJBU-GUBZILKMSA-N Arg-Asp-Met Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CCSC)C(O)=O RRGPUNYIPJXJBU-GUBZILKMSA-N 0.000 description 1
- TTXYKSADPSNOIF-IHRRRGAJSA-N Arg-Asp-Phe Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O TTXYKSADPSNOIF-IHRRRGAJSA-N 0.000 description 1
- JCAISGGAOQXEHJ-ZPFDUUQYSA-N Arg-Gln-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CCCN=C(N)N)N JCAISGGAOQXEHJ-ZPFDUUQYSA-N 0.000 description 1
- QAODJPUKWNNNRP-DCAQKATOSA-N Arg-Glu-Arg Chemical compound NC(N)=NCCC[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCN=C(N)N)C(O)=O QAODJPUKWNNNRP-DCAQKATOSA-N 0.000 description 1
- RKRSYHCNPFGMTA-CIUDSAMLSA-N Arg-Glu-Asn Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(N)=O)C(O)=O RKRSYHCNPFGMTA-CIUDSAMLSA-N 0.000 description 1
- MZRBYBIQTIKERR-GUBZILKMSA-N Arg-Glu-Gln Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCC(N)=O)C(O)=O MZRBYBIQTIKERR-GUBZILKMSA-N 0.000 description 1
- GOWZVQXTHUCNSQ-NHCYSSNCSA-N Arg-Glu-Val Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](C(C)C)C(O)=O GOWZVQXTHUCNSQ-NHCYSSNCSA-N 0.000 description 1
- RKQRHMKFNBYOTN-IHRRRGAJSA-N Arg-His-Lys Chemical compound C1=C(NC=N1)C[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CCCN=C(N)N)N RKQRHMKFNBYOTN-IHRRRGAJSA-N 0.000 description 1
- DNUKXVMPARLPFN-XUXIUFHCSA-N Arg-Leu-Ile Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O DNUKXVMPARLPFN-XUXIUFHCSA-N 0.000 description 1
- COXMUHNBYCVVRG-DCAQKATOSA-N Arg-Leu-Ser Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(O)=O COXMUHNBYCVVRG-DCAQKATOSA-N 0.000 description 1
- FSNVAJOPUDVQAR-AVGNSLFASA-N Arg-Lys-Arg Chemical compound NC(=N)NCCC[C@H](N)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O FSNVAJOPUDVQAR-AVGNSLFASA-N 0.000 description 1
- MJINRRBEMOLJAK-DCAQKATOSA-N Arg-Lys-Asp Chemical compound OC(=O)C[C@@H](C(O)=O)NC(=O)[C@H](CCCCN)NC(=O)[C@@H](N)CCCN=C(N)N MJINRRBEMOLJAK-DCAQKATOSA-N 0.000 description 1
- GRRXPUAICOGISM-RWMBFGLXSA-N Arg-Lys-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CCCCN)NC(=O)[C@H](CCCN=C(N)N)N)C(=O)O GRRXPUAICOGISM-RWMBFGLXSA-N 0.000 description 1
- PAPSMOYMQDWIOR-AVGNSLFASA-N Arg-Lys-Val Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](C(C)C)C(O)=O PAPSMOYMQDWIOR-AVGNSLFASA-N 0.000 description 1
- PYZPXCZNQSEHDT-GUBZILKMSA-N Arg-Met-Asn Chemical compound CSCC[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)O)NC(=O)[C@H](CCCN=C(N)N)N PYZPXCZNQSEHDT-GUBZILKMSA-N 0.000 description 1
- CZUHPNLXLWMYMG-UBHSHLNASA-N Arg-Phe-Ala Chemical compound NC(N)=NCCC[C@H](N)C(=O)N[C@H](C(=O)N[C@@H](C)C(O)=O)CC1=CC=CC=C1 CZUHPNLXLWMYMG-UBHSHLNASA-N 0.000 description 1
- YTMKMRSYXHBGER-IHRRRGAJSA-N Arg-Phe-Asn Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)O)NC(=O)[C@H](CCCN=C(N)N)N YTMKMRSYXHBGER-IHRRRGAJSA-N 0.000 description 1
- NGYHSXDNNOFHNE-AVGNSLFASA-N Arg-Pro-Leu Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CC(C)C)C(O)=O NGYHSXDNNOFHNE-AVGNSLFASA-N 0.000 description 1
- FRBAHXABMQXSJQ-FXQIFTODSA-N Arg-Ser-Ser Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CO)C(=O)N[C@@H](CO)C(O)=O FRBAHXABMQXSJQ-FXQIFTODSA-N 0.000 description 1
- AOJYORNRFWWEIV-IHRRRGAJSA-N Arg-Tyr-Asp Chemical compound NC(N)=NCCC[C@H](N)C(=O)N[C@H](C(=O)N[C@@H](CC(O)=O)C(O)=O)CC1=CC=C(O)C=C1 AOJYORNRFWWEIV-IHRRRGAJSA-N 0.000 description 1
- FMYQECOAIFGQGU-CYDGBPFRSA-N Arg-Val-Ile Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O FMYQECOAIFGQGU-CYDGBPFRSA-N 0.000 description 1
- XWGJDUSDTRPQRK-ZLUOBGJFSA-N Asn-Ala-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@H](C)NC(=O)[C@@H](N)CC(N)=O XWGJDUSDTRPQRK-ZLUOBGJFSA-N 0.000 description 1
- MEFGKQUUYZOLHM-GMOBBJLQSA-N Asn-Arg-Ile Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O MEFGKQUUYZOLHM-GMOBBJLQSA-N 0.000 description 1
- WPOLSNAQGVHROR-GUBZILKMSA-N Asn-Gln-Leu Chemical compound CC(C)C[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CC(=O)N)N WPOLSNAQGVHROR-GUBZILKMSA-N 0.000 description 1
- HCAUEJAQCXVQQM-ACZMJKKPSA-N Asn-Glu-Asp Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(O)=O)C(O)=O HCAUEJAQCXVQQM-ACZMJKKPSA-N 0.000 description 1
- QYXNFROWLZPWPC-FXQIFTODSA-N Asn-Glu-Gln Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCC(N)=O)C(O)=O QYXNFROWLZPWPC-FXQIFTODSA-N 0.000 description 1
- BZMWJLLUAKSIMH-FXQIFTODSA-N Asn-Glu-Glu Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCC(O)=O)C(O)=O BZMWJLLUAKSIMH-FXQIFTODSA-N 0.000 description 1
- FTCGGKNCJZOPNB-WHFBIAKZSA-N Asn-Gly-Ser Chemical compound NC(=O)C[C@H](N)C(=O)NCC(=O)N[C@@H](CO)C(O)=O FTCGGKNCJZOPNB-WHFBIAKZSA-N 0.000 description 1
- SPCONPVIDFMDJI-QSFUFRPTSA-N Asn-Ile-Val Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](C(C)C)C(O)=O SPCONPVIDFMDJI-QSFUFRPTSA-N 0.000 description 1
- DJIMLSXHXKWADV-CIUDSAMLSA-N Asn-Leu-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@@H](N)CC(N)=O DJIMLSXHXKWADV-CIUDSAMLSA-N 0.000 description 1
- LSJQOMAZIKQMTJ-SRVKXCTJSA-N Asn-Phe-Asp Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CC(O)=O)C(O)=O LSJQOMAZIKQMTJ-SRVKXCTJSA-N 0.000 description 1
- RAUPFUCUDBQYHE-AVGNSLFASA-N Asn-Phe-Glu Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CCC(O)=O)C(O)=O RAUPFUCUDBQYHE-AVGNSLFASA-N 0.000 description 1
- HZZIFFOVHLWGCS-KKUMJFAQSA-N Asn-Phe-Leu Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CC(C)C)C(O)=O HZZIFFOVHLWGCS-KKUMJFAQSA-N 0.000 description 1
- YUUIAUXBNOHFRJ-IHRRRGAJSA-N Asn-Phe-Met Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CCSC)C(O)=O YUUIAUXBNOHFRJ-IHRRRGAJSA-N 0.000 description 1
- KTDWFWNZLLFEFU-KKUMJFAQSA-N Asn-Tyr-His Chemical compound C1=CC(=CC=C1C[C@@H](C(=O)N[C@@H](CC2=CN=CN2)C(=O)O)NC(=O)[C@H](CC(=O)N)N)O KTDWFWNZLLFEFU-KKUMJFAQSA-N 0.000 description 1
- CGYKCTPUGXFPMG-IHPCNDPISA-N Asn-Tyr-Trp Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(O)=O CGYKCTPUGXFPMG-IHPCNDPISA-N 0.000 description 1
- XBQSLMACWDXWLJ-GHCJXIJMSA-N Asp-Ala-Ile Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](C)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O XBQSLMACWDXWLJ-GHCJXIJMSA-N 0.000 description 1
- BLQBMRNMBAYREH-UWJYBYFXSA-N Asp-Ala-Tyr Chemical compound N[C@@H](CC(O)=O)C(=O)N[C@@H](C)C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)O BLQBMRNMBAYREH-UWJYBYFXSA-N 0.000 description 1
- WSOKZUVWBXVJHX-CIUDSAMLSA-N Asp-Arg-Glu Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(O)=O)C(O)=O WSOKZUVWBXVJHX-CIUDSAMLSA-N 0.000 description 1
- MFMJRYHVLLEMQM-DCAQKATOSA-N Asp-Arg-His Chemical compound C1=C(NC=N1)C[C@@H](C(=O)O)NC(=O)[C@H](CCCN=C(N)N)NC(=O)[C@H](CC(=O)O)N MFMJRYHVLLEMQM-DCAQKATOSA-N 0.000 description 1
- SDHFVYLZFBDSQT-DCAQKATOSA-N Asp-Arg-Lys Chemical compound C(CCN)C[C@@H](C(=O)O)NC(=O)[C@H](CCCN=C(N)N)NC(=O)[C@H](CC(=O)O)N SDHFVYLZFBDSQT-DCAQKATOSA-N 0.000 description 1
- UGKZHCBLMLSANF-CIUDSAMLSA-N Asp-Asn-Leu Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CC(C)C)C(O)=O UGKZHCBLMLSANF-CIUDSAMLSA-N 0.000 description 1
- KNMRXHIAVXHCLW-ZLUOBGJFSA-N Asp-Asn-Ser Chemical compound C([C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H](CO)C(=O)O)N)C(=O)O KNMRXHIAVXHCLW-ZLUOBGJFSA-N 0.000 description 1
- RDRMWJBLOSRRAW-BYULHYEWSA-N Asp-Asn-Val Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](C(C)C)C(O)=O RDRMWJBLOSRRAW-BYULHYEWSA-N 0.000 description 1
- SBHUBSDEZQFJHJ-CIUDSAMLSA-N Asp-Asp-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CC(O)=O)NC(=O)[C@@H](N)CC(O)=O SBHUBSDEZQFJHJ-CIUDSAMLSA-N 0.000 description 1
- CELPEWWLSXMVPH-CIUDSAMLSA-N Asp-Asp-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@H](CC(O)=O)NC(=O)[C@@H](N)CC(O)=O CELPEWWLSXMVPH-CIUDSAMLSA-N 0.000 description 1
- QXHVOUSPVAWEMX-ZLUOBGJFSA-N Asp-Asp-Ser Chemical compound OC(=O)C[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CO)C(O)=O QXHVOUSPVAWEMX-ZLUOBGJFSA-N 0.000 description 1
- DXQOQMCLWWADMU-ACZMJKKPSA-N Asp-Gln-Ser Chemical compound OC(=O)C[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CO)C(O)=O DXQOQMCLWWADMU-ACZMJKKPSA-N 0.000 description 1
- VILLWIDTHYPSLC-PEFMBERDSA-N Asp-Glu-Ile Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O VILLWIDTHYPSLC-PEFMBERDSA-N 0.000 description 1
- RRKCPMGSRIDLNC-AVGNSLFASA-N Asp-Glu-Tyr Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O RRKCPMGSRIDLNC-AVGNSLFASA-N 0.000 description 1
- OMMIEVATLAGRCK-BYPYZUCNSA-N Asp-Gly-Gly Chemical compound OC(=O)C[C@H](N)C(=O)NCC(=O)NCC(O)=O OMMIEVATLAGRCK-BYPYZUCNSA-N 0.000 description 1
- TVIZQBFURPLQDV-DJFWLOJKSA-N Asp-His-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H](CC1=CN=CN1)NC(=O)[C@H](CC(=O)O)N TVIZQBFURPLQDV-DJFWLOJKSA-N 0.000 description 1
- CYCKJEFVFNRWEZ-UGYAYLCHSA-N Asp-Ile-Asn Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC(N)=O)C(O)=O CYCKJEFVFNRWEZ-UGYAYLCHSA-N 0.000 description 1
- SEMWSADZTMJELF-BYULHYEWSA-N Asp-Ile-Gly Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)CC)C(=O)NCC(O)=O SEMWSADZTMJELF-BYULHYEWSA-N 0.000 description 1
- KYQNAIMCTRZLNP-QSFUFRPTSA-N Asp-Ile-Val Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](C(C)C)C(O)=O KYQNAIMCTRZLNP-QSFUFRPTSA-N 0.000 description 1
- CLUMZOKVGUWUFD-CIUDSAMLSA-N Asp-Leu-Asn Chemical compound OC(=O)C[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC(N)=O)C(O)=O CLUMZOKVGUWUFD-CIUDSAMLSA-N 0.000 description 1
- ORRJQLIATJDMQM-HJGDQZAQSA-N Asp-Leu-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@@H](N)CC(O)=O ORRJQLIATJDMQM-HJGDQZAQSA-N 0.000 description 1
- XWSIYTYNLKCLJB-CIUDSAMLSA-N Asp-Lys-Asn Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(N)=O)C(O)=O XWSIYTYNLKCLJB-CIUDSAMLSA-N 0.000 description 1
- LBOVBQONZJRWPV-YUMQZZPRSA-N Asp-Lys-Gly Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CCCCN)C(=O)NCC(O)=O LBOVBQONZJRWPV-YUMQZZPRSA-N 0.000 description 1
- GKWFMNNNYZHJHV-SRVKXCTJSA-N Asp-Lys-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CCCCN)NC(=O)[C@@H](N)CC(O)=O GKWFMNNNYZHJHV-SRVKXCTJSA-N 0.000 description 1
- MYLZFUMPZCPJCJ-NHCYSSNCSA-N Asp-Lys-Val Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](C(C)C)C(O)=O MYLZFUMPZCPJCJ-NHCYSSNCSA-N 0.000 description 1
- BJDHEININLSZOT-KKUMJFAQSA-N Asp-Tyr-Lys Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CCCCN)C(O)=O BJDHEININLSZOT-KKUMJFAQSA-N 0.000 description 1
- SQIARYGNVQWOSB-BZSNNMDCSA-N Asp-Tyr-Phe Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O SQIARYGNVQWOSB-BZSNNMDCSA-N 0.000 description 1
- WAEDSQFVZJUHLI-BYULHYEWSA-N Asp-Val-Asp Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CC(O)=O)C(O)=O WAEDSQFVZJUHLI-BYULHYEWSA-N 0.000 description 1
- 241000589941 Azospirillum Species 0.000 description 1
- 101150023320 B16R gene Proteins 0.000 description 1
- 241000257169 Bacillus cereus ATCC 10987 Species 0.000 description 1
- 241000606124 Bacteroides fragilis Species 0.000 description 1
- 201000005943 Barth syndrome Diseases 0.000 description 1
- 241000586987 Bifidobacterium dentium Bd1 Species 0.000 description 1
- 241001209261 Bifidobacterium longum DJO10A Species 0.000 description 1
- 241000589173 Bradyrhizobium Species 0.000 description 1
- 238000010453 CRISPR/Cas method Methods 0.000 description 1
- AQGNHMOJWBZFQQ-UHFFFAOYSA-N CT 99021 Chemical compound CC1=CNC(C=2C(=NC(NCCNC=3N=CC(=CC=3)C#N)=NC=2)C=2C(=CC(Cl)=CC=2)Cl)=N1 AQGNHMOJWBZFQQ-UHFFFAOYSA-N 0.000 description 1
- 241001453247 Campylobacter jejuni subsp. doylei Species 0.000 description 1
- 241000941427 Campylobacter lari RM2100 Species 0.000 description 1
- 241001034636 Capnocytophaga ochracea DSM 7271 Species 0.000 description 1
- 241001112695 Clostridiales Species 0.000 description 1
- 241001509423 Clostridium botulinum B Species 0.000 description 1
- 241001509504 Clostridium botulinum F Species 0.000 description 1
- 108700010070 Codon Usage Proteins 0.000 description 1
- 108060005980 Collagenase Proteins 0.000 description 1
- 102000029816 Collagenase Human genes 0.000 description 1
- 241000186216 Corynebacterium Species 0.000 description 1
- 241000186227 Corynebacterium diphtheriae Species 0.000 description 1
- 241001644925 Corynebacterium efficiens Species 0.000 description 1
- 241001485655 Corynebacterium glutamicum ATCC 13032 Species 0.000 description 1
- 241000671338 Corynebacterium glutamicum R Species 0.000 description 1
- 241001525611 Corynebacterium kroppenstedtii DSM 44385 Species 0.000 description 1
- 230000005778 DNA damage Effects 0.000 description 1
- 231100000277 DNA damage Toxicity 0.000 description 1
- 230000006820 DNA synthesis Effects 0.000 description 1
- 241000252212 Danio rerio Species 0.000 description 1
- 102000016911 Deoxyribonucleases Human genes 0.000 description 1
- 108010053770 Deoxyribonucleases Proteins 0.000 description 1
- 241001082278 Desulfovibrio salexigens DSM 2638 Species 0.000 description 1
- 241000688137 Diaphorobacter Species 0.000 description 1
- 241000933091 Dinoroseobacter shibae DFL 12 = DSM 16493 Species 0.000 description 1
- 108010067770 Endopeptidase K Proteins 0.000 description 1
- 241000588724 Escherichia coli Species 0.000 description 1
- 241000206602 Eukaryota Species 0.000 description 1
- 241000608038 Fibrobacter succinogenes subsp. succinogenes S85 Species 0.000 description 1
- 108090000379 Fibroblast growth factor 2 Proteins 0.000 description 1
- 102000003974 Fibroblast growth factor 2 Human genes 0.000 description 1
- 102100037362 Fibronectin Human genes 0.000 description 1
- 108010067306 Fibronectins Proteins 0.000 description 1
- 241000359186 Finegoldia magna ATCC 29328 Species 0.000 description 1
- 108091092584 GDNA Proteins 0.000 description 1
- 101150106478 GPS1 gene Proteins 0.000 description 1
- KVYVOGYEMPEXBT-GUBZILKMSA-N Gln-Ala-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](C)NC(=O)[C@@H](N)CCC(N)=O KVYVOGYEMPEXBT-GUBZILKMSA-N 0.000 description 1
- PHZYLYASFWHLHJ-FXQIFTODSA-N Gln-Asn-Glu Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CCC(O)=O)C(O)=O PHZYLYASFWHLHJ-FXQIFTODSA-N 0.000 description 1
- ZPDVKYLJTOFQJV-WDSKDSINSA-N Gln-Asn-Gly Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(N)=O)C(=O)NCC(O)=O ZPDVKYLJTOFQJV-WDSKDSINSA-N 0.000 description 1
- KVXVVDFOZNYYKZ-DCAQKATOSA-N Gln-Gln-Leu Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(C)C)C(O)=O KVXVVDFOZNYYKZ-DCAQKATOSA-N 0.000 description 1
- SNLOOPZHAQDMJG-CIUDSAMLSA-N Gln-Glu-Glu Chemical compound NC(=O)CC[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCC(O)=O)C(O)=O SNLOOPZHAQDMJG-CIUDSAMLSA-N 0.000 description 1
- KDXKFBSNIJYNNR-YVNDNENWSA-N Gln-Glu-Ile Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O KDXKFBSNIJYNNR-YVNDNENWSA-N 0.000 description 1
- PNENQZWRFMUZOM-DCAQKATOSA-N Gln-Glu-Leu Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(O)=O PNENQZWRFMUZOM-DCAQKATOSA-N 0.000 description 1
- NNXIQPMZGZUFJJ-AVGNSLFASA-N Gln-His-Lys Chemical compound C1=C(NC=N1)C[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CCC(=O)N)N NNXIQPMZGZUFJJ-AVGNSLFASA-N 0.000 description 1
- GIVHPCWYVWUUSG-HVTMNAMFSA-N Gln-Ile-His Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)NC(=O)[C@H](CCC(=O)N)N GIVHPCWYVWUUSG-HVTMNAMFSA-N 0.000 description 1
- FTIJVMLAGRAYMJ-MNXVOIDGSA-N Gln-Ile-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H]([C@@H](C)CC)NC(=O)[C@@H](N)CCC(N)=O FTIJVMLAGRAYMJ-MNXVOIDGSA-N 0.000 description 1
- PSERKXGRRADTKA-MNXVOIDGSA-N Gln-Leu-Ile Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O PSERKXGRRADTKA-MNXVOIDGSA-N 0.000 description 1
- IULKWYSYZSURJK-AVGNSLFASA-N Gln-Leu-Lys Chemical compound NC(=O)CC[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(O)=O IULKWYSYZSURJK-AVGNSLFASA-N 0.000 description 1
- YPMDZWPZFOZYFG-GUBZILKMSA-N Gln-Leu-Ser Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(O)=O YPMDZWPZFOZYFG-GUBZILKMSA-N 0.000 description 1
- ZGHMRONFHDVXEF-AVGNSLFASA-N Gln-Ser-Phe Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O ZGHMRONFHDVXEF-AVGNSLFASA-N 0.000 description 1
- WTJIWXMJESRHMM-XDTLVQLUSA-N Gln-Tyr-Ala Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](C)C(O)=O WTJIWXMJESRHMM-XDTLVQLUSA-N 0.000 description 1
- JJKKWYQVHRUSDG-GUBZILKMSA-N Glu-Ala-Lys Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](C)C(=O)N[C@@H](CCCCN)C(O)=O JJKKWYQVHRUSDG-GUBZILKMSA-N 0.000 description 1
- KKCUFHUTMKQQCF-SRVKXCTJSA-N Glu-Arg-Leu Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(C)C)C(O)=O KKCUFHUTMKQQCF-SRVKXCTJSA-N 0.000 description 1
- YKLNMGJYMNPBCP-ACZMJKKPSA-N Glu-Asn-Asp Chemical compound C(CC(=O)O)[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H](CC(=O)O)C(=O)O)N YKLNMGJYMNPBCP-ACZMJKKPSA-N 0.000 description 1
- CKRUHITYRFNUKW-WDSKDSINSA-N Glu-Asn-Gly Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(N)=O)C(=O)NCC(O)=O CKRUHITYRFNUKW-WDSKDSINSA-N 0.000 description 1
- HJIFPJUEOGZWRI-GUBZILKMSA-N Glu-Asp-Lys Chemical compound C(CCN)C[C@@H](C(=O)O)NC(=O)[C@H](CC(=O)O)NC(=O)[C@H](CCC(=O)O)N HJIFPJUEOGZWRI-GUBZILKMSA-N 0.000 description 1
- PAQUJCSYVIBPLC-AVGNSLFASA-N Glu-Asp-Phe Chemical compound OC(=O)CC[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 PAQUJCSYVIBPLC-AVGNSLFASA-N 0.000 description 1
- ZXQPJYWZSFGWJB-AVGNSLFASA-N Glu-Cys-Phe Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)O)NC(=O)[C@H](CS)NC(=O)[C@H](CCC(=O)O)N ZXQPJYWZSFGWJB-AVGNSLFASA-N 0.000 description 1
- SJPMNHCEWPTRBR-BQBZGAKWSA-N Glu-Glu-Gly Chemical compound OC(=O)CC[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(=O)NCC(O)=O SJPMNHCEWPTRBR-BQBZGAKWSA-N 0.000 description 1
- VOORMNJKNBGYGK-YUMQZZPRSA-N Glu-Gly-Met Chemical compound CSCC[C@@H](C(=O)O)NC(=O)CNC(=O)[C@H](CCC(=O)O)N VOORMNJKNBGYGK-YUMQZZPRSA-N 0.000 description 1
- XOIATPHFYVWFEU-DCAQKATOSA-N Glu-His-Gln Chemical compound C1=C(NC=N1)C[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)O)NC(=O)[C@H](CCC(=O)O)N XOIATPHFYVWFEU-DCAQKATOSA-N 0.000 description 1
- CXRWMMRLEMVSEH-PEFMBERDSA-N Glu-Ile-Asn Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC(N)=O)C(O)=O CXRWMMRLEMVSEH-PEFMBERDSA-N 0.000 description 1
- WTMZXOPHTIVFCP-QEWYBTABSA-N Glu-Ile-Phe Chemical compound OC(=O)CC[C@H](N)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 WTMZXOPHTIVFCP-QEWYBTABSA-N 0.000 description 1
- ZSWGJYOZWBHROQ-RWRJDSDZSA-N Glu-Ile-Thr Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H]([C@@H](C)O)C(O)=O ZSWGJYOZWBHROQ-RWRJDSDZSA-N 0.000 description 1
- HVYWQYLBVXMXSV-GUBZILKMSA-N Glu-Leu-Ala Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](C)C(O)=O HVYWQYLBVXMXSV-GUBZILKMSA-N 0.000 description 1
- DNPCBMNFQVTHMA-DCAQKATOSA-N Glu-Leu-Gln Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(N)=O)C(O)=O DNPCBMNFQVTHMA-DCAQKATOSA-N 0.000 description 1
- NJCALAAIGREHDR-WDCWCFNPSA-N Glu-Leu-Thr Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O NJCALAAIGREHDR-WDCWCFNPSA-N 0.000 description 1
- ILWHFUZZCFYSKT-AVGNSLFASA-N Glu-Lys-Leu Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(C)C)C(O)=O ILWHFUZZCFYSKT-AVGNSLFASA-N 0.000 description 1
- ZGEJRLJEAMPEDV-SRVKXCTJSA-N Glu-Lys-Met Chemical compound CSCC[C@@H](C(=O)O)NC(=O)[C@H](CCCCN)NC(=O)[C@H](CCC(=O)O)N ZGEJRLJEAMPEDV-SRVKXCTJSA-N 0.000 description 1
- ZQYZDDXTNQXUJH-CIUDSAMLSA-N Glu-Met-Ala Chemical compound C[C@@H](C(=O)O)NC(=O)[C@H](CCSC)NC(=O)[C@H](CCC(=O)O)N ZQYZDDXTNQXUJH-CIUDSAMLSA-N 0.000 description 1
- SYAYROHMAIHWFB-KBIXCLLPSA-N Glu-Ser-Ile Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CO)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O SYAYROHMAIHWFB-KBIXCLLPSA-N 0.000 description 1
- BXSZPACYCMNKLS-AVGNSLFASA-N Glu-Ser-Phe Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O BXSZPACYCMNKLS-AVGNSLFASA-N 0.000 description 1
- TWYSSILQABLLME-HJGDQZAQSA-N Glu-Thr-Arg Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O TWYSSILQABLLME-HJGDQZAQSA-N 0.000 description 1
- RGJKYNUINKGPJN-RWRJDSDZSA-N Glu-Thr-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H]([C@@H](C)O)NC(=O)[C@H](CCC(=O)O)N RGJKYNUINKGPJN-RWRJDSDZSA-N 0.000 description 1
- KIEICAOUSNYOLM-NRPADANISA-N Glu-Val-Ala Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](C)C(O)=O KIEICAOUSNYOLM-NRPADANISA-N 0.000 description 1
- YQPFCZVKMUVZIN-AUTRQRHGSA-N Glu-Val-Gln Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(N)=O)C(O)=O YQPFCZVKMUVZIN-AUTRQRHGSA-N 0.000 description 1
- ZYRXTRTUCAVNBQ-GVXVVHGQSA-N Glu-Val-Lys Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CCC(=O)O)N ZYRXTRTUCAVNBQ-GVXVVHGQSA-N 0.000 description 1
- 241000032681 Gluconacetobacter Species 0.000 description 1
- 241000423296 Gluconacetobacter diazotrophicus PA1 5 Species 0.000 description 1
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 1
- VSVZIEVNUYDAFR-YUMQZZPRSA-N Gly-Ala-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](C)NC(=O)CN VSVZIEVNUYDAFR-YUMQZZPRSA-N 0.000 description 1
- LJPIRKICOISLKN-WHFBIAKZSA-N Gly-Ala-Ser Chemical compound NCC(=O)N[C@@H](C)C(=O)N[C@@H](CO)C(O)=O LJPIRKICOISLKN-WHFBIAKZSA-N 0.000 description 1
- OVSKVOOUFAKODB-UWVGGRQHSA-N Gly-Arg-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)CN)CCCN=C(N)N OVSKVOOUFAKODB-UWVGGRQHSA-N 0.000 description 1
- XCLCVBYNGXEVDU-WHFBIAKZSA-N Gly-Asn-Ser Chemical compound NCC(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CO)C(O)=O XCLCVBYNGXEVDU-WHFBIAKZSA-N 0.000 description 1
- FMNHBTKMRFVGRO-FOHZUACHSA-N Gly-Asn-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)[C@H](CC(N)=O)NC(=O)CN FMNHBTKMRFVGRO-FOHZUACHSA-N 0.000 description 1
- LCNXZQROPKFGQK-WHFBIAKZSA-N Gly-Asp-Ser Chemical compound NCC(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CO)C(O)=O LCNXZQROPKFGQK-WHFBIAKZSA-N 0.000 description 1
- YYPFZVIXAVDHIK-IUCAKERBSA-N Gly-Glu-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)CN YYPFZVIXAVDHIK-IUCAKERBSA-N 0.000 description 1
- CCQOOWAONKGYKQ-BYPYZUCNSA-N Gly-Gly-Ala Chemical compound OC(=O)[C@H](C)NC(=O)CNC(=O)CN CCQOOWAONKGYKQ-BYPYZUCNSA-N 0.000 description 1
- UFPXDFOYHVEIPI-BYPYZUCNSA-N Gly-Gly-Asp Chemical compound NCC(=O)NCC(=O)N[C@H](C(O)=O)CC(O)=O UFPXDFOYHVEIPI-BYPYZUCNSA-N 0.000 description 1
- XPJBQTCXPJNIFE-ZETCQYMHSA-N Gly-Gly-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)CNC(=O)CN XPJBQTCXPJNIFE-ZETCQYMHSA-N 0.000 description 1
- QITBQGJOXQYMOA-ZETCQYMHSA-N Gly-Gly-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)CNC(=O)CN QITBQGJOXQYMOA-ZETCQYMHSA-N 0.000 description 1
- SXJHOPPTOJACOA-QXEWZRGKSA-N Gly-Ile-Arg Chemical compound NCC(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@H](C(O)=O)CCCN=C(N)N SXJHOPPTOJACOA-QXEWZRGKSA-N 0.000 description 1
- SCWYHUQOOFRVHP-MBLNEYKQSA-N Gly-Ile-Thr Chemical compound NCC(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H]([C@@H](C)O)C(O)=O SCWYHUQOOFRVHP-MBLNEYKQSA-N 0.000 description 1
- AFWYPMDMDYCKMD-KBPBESRZSA-N Gly-Leu-Tyr Chemical compound NCC(=O)N[C@@H](CC(C)C)C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 AFWYPMDMDYCKMD-KBPBESRZSA-N 0.000 description 1
- VBOBNHSVQKKTOT-YUMQZZPRSA-N Gly-Lys-Ala Chemical compound [H]NCC(=O)N[C@@H](CCCCN)C(=O)N[C@@H](C)C(O)=O VBOBNHSVQKKTOT-YUMQZZPRSA-N 0.000 description 1
- FXGRXIATVXUAHO-WEDXCCLWSA-N Gly-Lys-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)CN)CCCCN FXGRXIATVXUAHO-WEDXCCLWSA-N 0.000 description 1
- GAFKBWKVXNERFA-QWRGUYRKSA-N Gly-Phe-Asp Chemical compound OC(=O)C[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)CN)CC1=CC=CC=C1 GAFKBWKVXNERFA-QWRGUYRKSA-N 0.000 description 1
- DBUNZBWUWCIELX-JHEQGTHGSA-N Gly-Thr-Glu Chemical compound [H]NCC(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCC(O)=O)C(O)=O DBUNZBWUWCIELX-JHEQGTHGSA-N 0.000 description 1
- RCHFYMASWAZQQZ-ZANVPECISA-N Gly-Trp-Ala Chemical compound C1=CC=C2C(C[C@@H](C(=O)N[C@@H](C)C(O)=O)NC(=O)CN)=CNC2=C1 RCHFYMASWAZQQZ-ZANVPECISA-N 0.000 description 1
- PNUFMLXHOLFRLD-KBPBESRZSA-N Gly-Tyr-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)CN)CC1=CC=C(O)C=C1 PNUFMLXHOLFRLD-KBPBESRZSA-N 0.000 description 1
- DNVDEMWIYLVIQU-RCOVLWMOSA-N Gly-Val-Asp Chemical compound NCC(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CC(O)=O)C(O)=O DNVDEMWIYLVIQU-RCOVLWMOSA-N 0.000 description 1
- 101150109546 HDR gene Proteins 0.000 description 1
- 241001453258 Helicobacter hepaticus Species 0.000 description 1
- 229920000209 Hexadimethrine bromide Polymers 0.000 description 1
- IPIVXQQRZXEUGW-UWJYBYFXSA-N His-Ala-His Chemical compound C([C@H](N)C(=O)N[C@@H](C)C(=O)N[C@@H](CC=1NC=NC=1)C(O)=O)C1=CN=CN1 IPIVXQQRZXEUGW-UWJYBYFXSA-N 0.000 description 1
- XINDHUAGVGCNSF-QSFUFRPTSA-N His-Ala-Ile Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](C)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O XINDHUAGVGCNSF-QSFUFRPTSA-N 0.000 description 1
- SVHKVHBPTOMLTO-DCAQKATOSA-N His-Arg-Asp Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(O)=O)C(O)=O SVHKVHBPTOMLTO-DCAQKATOSA-N 0.000 description 1
- MVADCDSCFTXCBT-CIUDSAMLSA-N His-Asp-Asp Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC(O)=O)C(O)=O MVADCDSCFTXCBT-CIUDSAMLSA-N 0.000 description 1
- IMCHNUANCIGUKS-SRVKXCTJSA-N His-Glu-Arg Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O IMCHNUANCIGUKS-SRVKXCTJSA-N 0.000 description 1
- XMENRVZYPBKBIL-AVGNSLFASA-N His-Glu-His Chemical compound N[C@@H](Cc1cnc[nH]1)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](Cc1cnc[nH]1)C(O)=O XMENRVZYPBKBIL-AVGNSLFASA-N 0.000 description 1
- JCOSMKPAOYDKRO-AVGNSLFASA-N His-Glu-Lys Chemical compound C1=C(NC=N1)C[C@@H](C(=O)N[C@@H](CCC(=O)O)C(=O)N[C@@H](CCCCN)C(=O)O)N JCOSMKPAOYDKRO-AVGNSLFASA-N 0.000 description 1
- LYCVKHSJGDMDLM-LURJTMIESA-N His-Gly Chemical compound OC(=O)CNC(=O)[C@@H](N)CC1=CN=CN1 LYCVKHSJGDMDLM-LURJTMIESA-N 0.000 description 1
- ZSKJIISDJXJQPV-BZSNNMDCSA-N His-Leu-Phe Chemical compound C([C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC=1C=CC=CC=1)C(O)=O)C1=CN=CN1 ZSKJIISDJXJQPV-BZSNNMDCSA-N 0.000 description 1
- SVVULKPWDBIPCO-BZSNNMDCSA-N His-Phe-Leu Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CC(C)C)C(O)=O SVVULKPWDBIPCO-BZSNNMDCSA-N 0.000 description 1
- WCHONUZTYDQMBY-PYJNHQTQSA-N His-Pro-Ile Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)N1CCC[C@H]1C(=O)N[C@@H]([C@@H](C)CC)C(O)=O WCHONUZTYDQMBY-PYJNHQTQSA-N 0.000 description 1
- DAKSMIWQZPHRIB-BZSNNMDCSA-N His-Tyr-Leu Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(C)C)C(O)=O DAKSMIWQZPHRIB-BZSNNMDCSA-N 0.000 description 1
- 101000578059 Homo sapiens Non-homologous end-joining factor 1 Proteins 0.000 description 1
- 206010020751 Hypersensitivity Diseases 0.000 description 1
- LQSBBHNVAVNZSX-GHCJXIJMSA-N Ile-Ala-Asn Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](C)C(=O)N[C@@H](CC(=O)N)C(=O)O)N LQSBBHNVAVNZSX-GHCJXIJMSA-N 0.000 description 1
- YOTNPRLPIPHQSB-XUXIUFHCSA-N Ile-Arg-Lys Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CCCCN)C(=O)O)N YOTNPRLPIPHQSB-XUXIUFHCSA-N 0.000 description 1
- NBJAAWYRLGCJOF-UGYAYLCHSA-N Ile-Asp-Asn Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC(=O)O)C(=O)N[C@@H](CC(=O)N)C(=O)O)N NBJAAWYRLGCJOF-UGYAYLCHSA-N 0.000 description 1
- RGSOCXHDOPQREB-ZPFDUUQYSA-N Ile-Asp-Leu Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC(=O)O)C(=O)N[C@@H](CC(C)C)C(=O)O)N RGSOCXHDOPQREB-ZPFDUUQYSA-N 0.000 description 1
- KUHFPGIVBOCRMV-MNXVOIDGSA-N Ile-Gln-Leu Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)N[C@@H](CC(C)C)C(=O)O)N KUHFPGIVBOCRMV-MNXVOIDGSA-N 0.000 description 1
- LKACSKJPTFSBHR-MNXVOIDGSA-N Ile-Gln-Lys Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)N[C@@H](CCCCN)C(=O)O)N LKACSKJPTFSBHR-MNXVOIDGSA-N 0.000 description 1
- JDAWAWXGAUZPNJ-ZPFDUUQYSA-N Ile-Glu-Arg Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCC(=O)O)C(=O)N[C@@H](CCCN=C(N)N)C(=O)O)N JDAWAWXGAUZPNJ-ZPFDUUQYSA-N 0.000 description 1
- LGMUPVWZEYYUMU-YVNDNENWSA-N Ile-Glu-Gln Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCC(=O)O)C(=O)N[C@@H](CCC(=O)N)C(=O)O)N LGMUPVWZEYYUMU-YVNDNENWSA-N 0.000 description 1
- MTFVYKQRLXYAQN-LAEOZQHASA-N Ile-Glu-Gly Chemical compound [H]N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CCC(O)=O)C(=O)NCC(O)=O MTFVYKQRLXYAQN-LAEOZQHASA-N 0.000 description 1
- PNDMHTTXXPUQJH-RWRJDSDZSA-N Ile-Glu-Thr Chemical compound N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CCC(=O)O)C(=O)N[C@@H]([C@H](O)C)C(=O)O PNDMHTTXXPUQJH-RWRJDSDZSA-N 0.000 description 1
- KFVUBLZRFSVDGO-BYULHYEWSA-N Ile-Gly-Asp Chemical compound CC[C@H](C)[C@H](N)C(=O)NCC(=O)N[C@H](C(O)=O)CC(O)=O KFVUBLZRFSVDGO-BYULHYEWSA-N 0.000 description 1
- CDGLBYSAZFIIJO-RCOVLWMOSA-N Ile-Gly-Gly Chemical compound CC[C@H](C)[C@H]([NH3+])C(=O)NCC(=O)NCC([O-])=O CDGLBYSAZFIIJO-RCOVLWMOSA-N 0.000 description 1
- NYEYYMLUABXDMC-NHCYSSNCSA-N Ile-Gly-Leu Chemical compound CC[C@H](C)[C@@H](C(=O)NCC(=O)N[C@@H](CC(C)C)C(=O)O)N NYEYYMLUABXDMC-NHCYSSNCSA-N 0.000 description 1
- HYLIOBDWPQNLKI-HVTMNAMFSA-N Ile-His-Gln Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)N[C@@H](CCC(=O)N)C(=O)O)N HYLIOBDWPQNLKI-HVTMNAMFSA-N 0.000 description 1
- HUWYGQOISIJNMK-SIGLWIIPSA-N Ile-Ile-His Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)N HUWYGQOISIJNMK-SIGLWIIPSA-N 0.000 description 1
- CSQNHSGHAPRGPQ-YTFOTSKYSA-N Ile-Ile-Lys Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CCCCN)C(=O)O)N CSQNHSGHAPRGPQ-YTFOTSKYSA-N 0.000 description 1
- PKGGWLOLRLOPGK-XUXIUFHCSA-N Ile-Leu-Arg Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@H](C(O)=O)CCCN=C(N)N PKGGWLOLRLOPGK-XUXIUFHCSA-N 0.000 description 1
- OUUCIIJSBIBCHB-ZPFDUUQYSA-N Ile-Leu-Asp Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC(O)=O)C(O)=O OUUCIIJSBIBCHB-ZPFDUUQYSA-N 0.000 description 1
- YGDWPQCLFJNMOL-MNXVOIDGSA-N Ile-Leu-Gln Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(=O)N)C(=O)O)N YGDWPQCLFJNMOL-MNXVOIDGSA-N 0.000 description 1
- HUORUFRRJHELPD-MNXVOIDGSA-N Ile-Leu-Glu Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(=O)O)C(=O)O)N HUORUFRRJHELPD-MNXVOIDGSA-N 0.000 description 1
- ADDYYRVQQZFIMW-MNXVOIDGSA-N Ile-Lys-Glu Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCC(=O)O)C(=O)O)N ADDYYRVQQZFIMW-MNXVOIDGSA-N 0.000 description 1
- GVNNAHIRSDRIII-AJNGGQMLSA-N Ile-Lys-Lys Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCCCN)C(=O)O)N GVNNAHIRSDRIII-AJNGGQMLSA-N 0.000 description 1
- GLYJPWIRLBAIJH-UHFFFAOYSA-N Ile-Lys-Pro Natural products CCC(C)C(N)C(=O)NC(CCCCN)C(=O)N1CCCC1C(O)=O GLYJPWIRLBAIJH-UHFFFAOYSA-N 0.000 description 1
- RVNOXPZHMUWCLW-GMOBBJLQSA-N Ile-Met-Asn Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCSC)C(=O)N[C@@H](CC(=O)N)C(=O)O)N RVNOXPZHMUWCLW-GMOBBJLQSA-N 0.000 description 1
- RCMNUBZKIIJCOI-ZPFDUUQYSA-N Ile-Met-Glu Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCSC)C(=O)N[C@@H](CCC(=O)O)C(=O)O)N RCMNUBZKIIJCOI-ZPFDUUQYSA-N 0.000 description 1
- CIDLJWVDMNDKPT-FIRPJDEBSA-N Ile-Phe-Phe Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CC2=CC=CC=C2)C(=O)O)N CIDLJWVDMNDKPT-FIRPJDEBSA-N 0.000 description 1
- BJECXJHLUJXPJQ-PYJNHQTQSA-N Ile-Pro-His Chemical compound CC[C@H](C)[C@@H](C(=O)N1CCC[C@H]1C(=O)N[C@@H](CC2=CN=CN2)C(=O)O)N BJECXJHLUJXPJQ-PYJNHQTQSA-N 0.000 description 1
- ZNOBVZFCHNHKHA-KBIXCLLPSA-N Ile-Ser-Glu Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CCC(=O)O)C(=O)O)N ZNOBVZFCHNHKHA-KBIXCLLPSA-N 0.000 description 1
- ZLFNNVATRMCAKN-ZKWXMUAHSA-N Ile-Ser-Gly Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CO)C(=O)NCC(=O)O)N ZLFNNVATRMCAKN-ZKWXMUAHSA-N 0.000 description 1
- JSLIXOUMAOUGBN-JUKXBJQTSA-N Ile-Tyr-His Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)N[C@@H](CC2=CN=CN2)C(=O)O)N JSLIXOUMAOUGBN-JUKXBJQTSA-N 0.000 description 1
- DLEBSGAVWRPTIX-PEDHHIEDSA-N Ile-Val-Ile Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@H](C(O)=O)[C@@H](C)CC DLEBSGAVWRPTIX-PEDHHIEDSA-N 0.000 description 1
- SWNRZNLXMXRCJC-VKOGCVSHSA-N Ile-Val-Trp Chemical compound C1=CC=C2C(C[C@H](NC(=O)[C@H](C(C)C)NC(=O)[C@@H](N)[C@@H](C)CC)C(O)=O)=CNC2=C1 SWNRZNLXMXRCJC-VKOGCVSHSA-N 0.000 description 1
- 108010065920 Insulin Lispro Proteins 0.000 description 1
- 102100034343 Integrase Human genes 0.000 description 1
- 108010061833 Integrases Proteins 0.000 description 1
- 108010015268 Integration Host Factors Proteins 0.000 description 1
- 241001596092 Kribbella flavida DSM 17836 Species 0.000 description 1
- FADYJNXDPBKVCA-UHFFFAOYSA-N L-Phenylalanyl-L-lysin Natural products NCCCCC(C(O)=O)NC(=O)C(N)CC1=CC=CC=C1 FADYJNXDPBKVCA-UHFFFAOYSA-N 0.000 description 1
- LHSGPCFBGJHPCY-UHFFFAOYSA-N L-leucine-L-tyrosine Natural products CC(C)CC(N)C(=O)NC(C(O)=O)CC1=CC=C(O)C=C1 LHSGPCFBGJHPCY-UHFFFAOYSA-N 0.000 description 1
- SENJXOPIZNYLHU-UHFFFAOYSA-N L-leucyl-L-arginine Natural products CC(C)CC(N)C(=O)NC(C(O)=O)CCCN=C(N)N SENJXOPIZNYLHU-UHFFFAOYSA-N 0.000 description 1
- 244000199866 Lactobacillus casei Species 0.000 description 1
- 235000013958 Lactobacillus casei Nutrition 0.000 description 1
- 241000917009 Lactobacillus rhamnosus GG Species 0.000 description 1
- 241001427851 Lactobacillus salivarius UCC118 Species 0.000 description 1
- 241001193656 Legionella pneumophila str. Paris Species 0.000 description 1
- LJHGALIOHLRRQN-DCAQKATOSA-N Leu-Ala-Arg Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@H](C(O)=O)CCCN=C(N)N LJHGALIOHLRRQN-DCAQKATOSA-N 0.000 description 1
- ZRLUISBDKUWAIZ-CIUDSAMLSA-N Leu-Ala-Asp Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@H](C(O)=O)CC(O)=O ZRLUISBDKUWAIZ-CIUDSAMLSA-N 0.000 description 1
- MJOZZTKJZQFKDK-GUBZILKMSA-N Leu-Ala-Gln Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@H](C(O)=O)CCC(N)=O MJOZZTKJZQFKDK-GUBZILKMSA-N 0.000 description 1
- WNGVUZWBXZKQES-YUMQZZPRSA-N Leu-Ala-Gly Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](C)C(=O)NCC(O)=O WNGVUZWBXZKQES-YUMQZZPRSA-N 0.000 description 1
- PBCHMHROGNUXMK-DLOVCJGASA-N Leu-Ala-His Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@H](C(O)=O)CC1=CN=CN1 PBCHMHROGNUXMK-DLOVCJGASA-N 0.000 description 1
- YOZCKMXHBYKOMQ-IHRRRGAJSA-N Leu-Arg-Lys Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CCCCN)C(=O)O)N YOZCKMXHBYKOMQ-IHRRRGAJSA-N 0.000 description 1
- WGNOPSQMIQERPK-UHFFFAOYSA-N Leu-Asn-Pro Natural products CC(C)CC(N)C(=O)NC(CC(=O)N)C(=O)N1CCCC1C(=O)O WGNOPSQMIQERPK-UHFFFAOYSA-N 0.000 description 1
- DLCOFDAHNMMQPP-SRVKXCTJSA-N Leu-Asp-Leu Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC(C)C)C(O)=O DLCOFDAHNMMQPP-SRVKXCTJSA-N 0.000 description 1
- PVMPDMIKUVNOBD-CIUDSAMLSA-N Leu-Asp-Ser Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CO)C(O)=O PVMPDMIKUVNOBD-CIUDSAMLSA-N 0.000 description 1
- ZFNLIDNJUWNIJL-WDCWCFNPSA-N Leu-Glu-Thr Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O ZFNLIDNJUWNIJL-WDCWCFNPSA-N 0.000 description 1
- OXRLYTYUXAQTHP-YUMQZZPRSA-N Leu-Gly-Ala Chemical compound [H]N[C@@H](CC(C)C)C(=O)NCC(=O)N[C@@H](C)C(O)=O OXRLYTYUXAQTHP-YUMQZZPRSA-N 0.000 description 1
- VGPCJSXPPOQPBK-YUMQZZPRSA-N Leu-Gly-Ser Chemical compound CC(C)C[C@H](N)C(=O)NCC(=O)N[C@@H](CO)C(O)=O VGPCJSXPPOQPBK-YUMQZZPRSA-N 0.000 description 1
- USLNHQZCDQJBOV-ZPFDUUQYSA-N Leu-Ile-Asn Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC(N)=O)C(O)=O USLNHQZCDQJBOV-ZPFDUUQYSA-N 0.000 description 1
- HNDWYLYAYNBWMP-AJNGGQMLSA-N Leu-Ile-Lys Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CC(C)C)N HNDWYLYAYNBWMP-AJNGGQMLSA-N 0.000 description 1
- LXKNSJLSGPNHSK-KKUMJFAQSA-N Leu-Leu-Lys Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(=O)O)N LXKNSJLSGPNHSK-KKUMJFAQSA-N 0.000 description 1
- RXGLHDWAZQECBI-SRVKXCTJSA-N Leu-Leu-Ser Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(O)=O RXGLHDWAZQECBI-SRVKXCTJSA-N 0.000 description 1
- RZXLZBIUTDQHJQ-SRVKXCTJSA-N Leu-Lys-Asp Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(O)=O)C(O)=O RZXLZBIUTDQHJQ-SRVKXCTJSA-N 0.000 description 1
- HVHRPWQEQHIQJF-AVGNSLFASA-N Leu-Lys-Glu Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCC(O)=O)C(O)=O HVHRPWQEQHIQJF-AVGNSLFASA-N 0.000 description 1
- ZAVCJRJOQKIOJW-KKUMJFAQSA-N Leu-Phe-Asp Chemical compound CC(C)C[C@H](N)C(=O)N[C@H](C(=O)N[C@@H](CC(O)=O)C(O)=O)CC1=CC=CC=C1 ZAVCJRJOQKIOJW-KKUMJFAQSA-N 0.000 description 1
- AIRUUHAOKGVJAD-JYJNAYRXSA-N Leu-Phe-Glu Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CCC(O)=O)C(O)=O AIRUUHAOKGVJAD-JYJNAYRXSA-N 0.000 description 1
- DRWMRVFCKKXHCH-BZSNNMDCSA-N Leu-Phe-Leu Chemical compound CC(C)C[C@H]([NH3+])C(=O)N[C@H](C(=O)N[C@@H](CC(C)C)C([O-])=O)CC1=CC=CC=C1 DRWMRVFCKKXHCH-BZSNNMDCSA-N 0.000 description 1
- PJWOOBTYQNNRBF-BZSNNMDCSA-N Leu-Phe-Lys Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CCCCN)C(=O)O)N PJWOOBTYQNNRBF-BZSNNMDCSA-N 0.000 description 1
- YWKNKRAKOCLOLH-OEAJRASXSA-N Leu-Phe-Thr Chemical compound CC(C)C[C@H](N)C(=O)N[C@H](C(=O)N[C@@H]([C@@H](C)O)C(O)=O)CC1=CC=CC=C1 YWKNKRAKOCLOLH-OEAJRASXSA-N 0.000 description 1
- CHJKEDSZNSONPS-DCAQKATOSA-N Leu-Pro-Ser Chemical compound [H]N[C@@H](CC(C)C)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CO)C(O)=O CHJKEDSZNSONPS-DCAQKATOSA-N 0.000 description 1
- IZPVWNSAVUQBGP-CIUDSAMLSA-N Leu-Ser-Asp Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(O)=O)C(O)=O IZPVWNSAVUQBGP-CIUDSAMLSA-N 0.000 description 1
- GOFJOGXGMPHOGL-DCAQKATOSA-N Leu-Ser-Met Chemical compound CSCC[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CC(C)C GOFJOGXGMPHOGL-DCAQKATOSA-N 0.000 description 1
- AEDWWMMHUGYIFD-HJGDQZAQSA-N Leu-Thr-Asn Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(N)=O)C(O)=O AEDWWMMHUGYIFD-HJGDQZAQSA-N 0.000 description 1
- QWWPYKKLXWOITQ-VOAKCMCISA-N Leu-Thr-Leu Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@H](C(O)=O)CC(C)C QWWPYKKLXWOITQ-VOAKCMCISA-N 0.000 description 1
- DAYQSYGBCUKVKT-VOAKCMCISA-N Leu-Thr-Lys Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCCCN)C(O)=O DAYQSYGBCUKVKT-VOAKCMCISA-N 0.000 description 1
- KLSUAWUZBMAZCL-RHYQMDGZSA-N Leu-Thr-Pro Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N1CCC[C@H]1C(O)=O KLSUAWUZBMAZCL-RHYQMDGZSA-N 0.000 description 1
- VJGQRELPQWNURN-JYJNAYRXSA-N Leu-Tyr-Glu Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CCC(O)=O)C(O)=O VJGQRELPQWNURN-JYJNAYRXSA-N 0.000 description 1
- YIRIDPUGZKHMHT-ACRUOGEOSA-N Leu-Tyr-Tyr Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O YIRIDPUGZKHMHT-ACRUOGEOSA-N 0.000 description 1
- FBNPMTNBFFAMMH-AVGNSLFASA-N Leu-Val-Arg Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@H](C(O)=O)CCCN=C(N)N FBNPMTNBFFAMMH-AVGNSLFASA-N 0.000 description 1
- FBNPMTNBFFAMMH-UHFFFAOYSA-N Leu-Val-Arg Natural products CC(C)CC(N)C(=O)NC(C(C)C)C(=O)NC(C(O)=O)CCCN=C(N)N FBNPMTNBFFAMMH-UHFFFAOYSA-N 0.000 description 1
- AIMGJYMCTAABEN-GVXVVHGQSA-N Leu-Val-Glu Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(O)=O)C(O)=O AIMGJYMCTAABEN-GVXVVHGQSA-N 0.000 description 1
- YQFZRHYZLARWDY-IHRRRGAJSA-N Leu-Val-Lys Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@H](C(O)=O)CCCCN YQFZRHYZLARWDY-IHRRRGAJSA-N 0.000 description 1
- 239000012097 Lipofectamine 2000 Substances 0.000 description 1
- 241000186805 Listeria innocua Species 0.000 description 1
- MPGHETGWWWUHPY-CIUDSAMLSA-N Lys-Ala-Asp Chemical compound OC(=O)C[C@@H](C(O)=O)NC(=O)[C@H](C)NC(=O)[C@@H](N)CCCCN MPGHETGWWWUHPY-CIUDSAMLSA-N 0.000 description 1
- XFIHDSBIPWEYJJ-YUMQZZPRSA-N Lys-Ala-Gly Chemical compound OC(=O)CNC(=O)[C@H](C)NC(=O)[C@@H](N)CCCCN XFIHDSBIPWEYJJ-YUMQZZPRSA-N 0.000 description 1
- NFLFJGGKOHYZJF-BJDJZHNGSA-N Lys-Ala-Ile Chemical compound CC[C@H](C)[C@@H](C(O)=O)NC(=O)[C@H](C)NC(=O)[C@@H](N)CCCCN NFLFJGGKOHYZJF-BJDJZHNGSA-N 0.000 description 1
- IXHKPDJKKCUKHS-GARJFASQSA-N Lys-Ala-Pro Chemical compound C[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CCCCN)N IXHKPDJKKCUKHS-GARJFASQSA-N 0.000 description 1
- SWWCDAGDQHTKIE-RHYQMDGZSA-N Lys-Arg-Thr Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H]([C@@H](C)O)C(O)=O SWWCDAGDQHTKIE-RHYQMDGZSA-N 0.000 description 1
- HQVDJTYKCMIWJP-YUMQZZPRSA-N Lys-Asn-Gly Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC(N)=O)C(=O)NCC(O)=O HQVDJTYKCMIWJP-YUMQZZPRSA-N 0.000 description 1
- DGWXCIORNLWGGG-CIUDSAMLSA-N Lys-Asn-Ser Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CO)C(O)=O DGWXCIORNLWGGG-CIUDSAMLSA-N 0.000 description 1
- IWWMPCPLFXFBAF-SRVKXCTJSA-N Lys-Asp-Leu Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC(C)C)C(O)=O IWWMPCPLFXFBAF-SRVKXCTJSA-N 0.000 description 1
- LMVOVCYVZBBWQB-SRVKXCTJSA-N Lys-Asp-Lys Chemical compound NCCCC[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@H](C(O)=O)CCCCN LMVOVCYVZBBWQB-SRVKXCTJSA-N 0.000 description 1
- KWUKZRFFKPLUPE-HJGDQZAQSA-N Lys-Asp-Thr Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O KWUKZRFFKPLUPE-HJGDQZAQSA-N 0.000 description 1
- PHHYNOUOUWYQRO-XIRDDKMYSA-N Lys-Asp-Trp Chemical compound C1=CC=C2C(=C1)C(=CN2)C[C@@H](C(=O)O)NC(=O)[C@H](CC(=O)O)NC(=O)[C@H](CCCCN)N PHHYNOUOUWYQRO-XIRDDKMYSA-N 0.000 description 1
- WTZUSCUIVPVCRH-SRVKXCTJSA-N Lys-Gln-Arg Chemical compound NCCCC[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@H](C(O)=O)CCCN=C(N)N WTZUSCUIVPVCRH-SRVKXCTJSA-N 0.000 description 1
- RZHLIPMZXOEJTL-AVGNSLFASA-N Lys-Gln-Leu Chemical compound CC(C)C[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CCCCN)N RZHLIPMZXOEJTL-AVGNSLFASA-N 0.000 description 1
- NNCDAORZCMPZPX-GUBZILKMSA-N Lys-Gln-Ser Chemical compound C(CCN)C[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)N[C@@H](CO)C(=O)O)N NNCDAORZCMPZPX-GUBZILKMSA-N 0.000 description 1
- LLSUNJYOSCOOEB-GUBZILKMSA-N Lys-Glu-Asp Chemical compound NCCCC[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(O)=O)C(O)=O LLSUNJYOSCOOEB-GUBZILKMSA-N 0.000 description 1
- KZOHPCYVORJBLG-AVGNSLFASA-N Lys-Glu-His Chemical compound C1=C(NC=N1)C[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)O)NC(=O)[C@H](CCCCN)N KZOHPCYVORJBLG-AVGNSLFASA-N 0.000 description 1
- QZONCCHVHCOBSK-YUMQZZPRSA-N Lys-Gly-Asn Chemical compound [H]N[C@@H](CCCCN)C(=O)NCC(=O)N[C@@H](CC(N)=O)C(O)=O QZONCCHVHCOBSK-YUMQZZPRSA-N 0.000 description 1
- XNKDCYABMBBEKN-IUCAKERBSA-N Lys-Gly-Gln Chemical compound NCCCC[C@H](N)C(=O)NCC(=O)N[C@H](C(O)=O)CCC(N)=O XNKDCYABMBBEKN-IUCAKERBSA-N 0.000 description 1
- FHIAJWBDZVHLAH-YUMQZZPRSA-N Lys-Gly-Ser Chemical compound NCCCC[C@H](N)C(=O)NCC(=O)N[C@@H](CO)C(O)=O FHIAJWBDZVHLAH-YUMQZZPRSA-N 0.000 description 1
- SPCHLZUWJTYZFC-IHRRRGAJSA-N Lys-His-Val Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](C(C)C)C(O)=O SPCHLZUWJTYZFC-IHRRRGAJSA-N 0.000 description 1
- QBEPTBMRQALPEV-MNXVOIDGSA-N Lys-Ile-Glu Chemical compound OC(=O)CC[C@@H](C(O)=O)NC(=O)[C@H]([C@@H](C)CC)NC(=O)[C@@H](N)CCCCN QBEPTBMRQALPEV-MNXVOIDGSA-N 0.000 description 1
- JYXBNQOKPRQNQS-YTFOTSKYSA-N Lys-Ile-Ile Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O JYXBNQOKPRQNQS-YTFOTSKYSA-N 0.000 description 1
- ZXFRGTAIIZHNHG-AJNGGQMLSA-N Lys-Ile-Leu Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC(C)C)C(=O)O)NC(=O)[C@H](CCCCN)N ZXFRGTAIIZHNHG-AJNGGQMLSA-N 0.000 description 1
- SKRGVGLIRUGANF-AVGNSLFASA-N Lys-Leu-Glu Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(O)=O)C(O)=O SKRGVGLIRUGANF-AVGNSLFASA-N 0.000 description 1
- XIZQPFCRXLUNMK-BZSNNMDCSA-N Lys-Leu-Phe Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)O)NC(=O)[C@H](CCCCN)N XIZQPFCRXLUNMK-BZSNNMDCSA-N 0.000 description 1
- VUTWYNQUSJWBHO-BZSNNMDCSA-N Lys-Leu-Tyr Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O VUTWYNQUSJWBHO-BZSNNMDCSA-N 0.000 description 1
- XOQMURBBIXRRCR-SRVKXCTJSA-N Lys-Lys-Ala Chemical compound OC(=O)[C@H](C)NC(=O)[C@H](CCCCN)NC(=O)[C@@H](N)CCCCN XOQMURBBIXRRCR-SRVKXCTJSA-N 0.000 description 1
- GAHJXEMYXKLZRQ-AJNGGQMLSA-N Lys-Lys-Ile Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O GAHJXEMYXKLZRQ-AJNGGQMLSA-N 0.000 description 1
- HVAUKHLDSDDROB-KKUMJFAQSA-N Lys-Lys-Leu Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(C)C)C(O)=O HVAUKHLDSDDROB-KKUMJFAQSA-N 0.000 description 1
- PLDJDCJLRCYPJB-VOAKCMCISA-N Lys-Lys-Thr Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H]([C@@H](C)O)C(O)=O PLDJDCJLRCYPJB-VOAKCMCISA-N 0.000 description 1
- BXPHMHQHYHILBB-BZSNNMDCSA-N Lys-Lys-Tyr Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O BXPHMHQHYHILBB-BZSNNMDCSA-N 0.000 description 1
- MTBLFIQZECOEBY-IHRRRGAJSA-N Lys-Met-Lys Chemical compound NCCCC[C@H](N)C(=O)N[C@@H](CCSC)C(=O)N[C@@H](CCCCN)C(O)=O MTBLFIQZECOEBY-IHRRRGAJSA-N 0.000 description 1
- QBHGXFQJFPWJIH-XUXIUFHCSA-N Lys-Pro-Ile Chemical compound CC[C@H](C)[C@@H](C(O)=O)NC(=O)[C@@H]1CCCN1C(=O)[C@@H](N)CCCCN QBHGXFQJFPWJIH-XUXIUFHCSA-N 0.000 description 1
- WQDKIVRHTQYJSN-DCAQKATOSA-N Lys-Ser-Arg Chemical compound C(CCN)C[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CCCN=C(N)N)C(=O)O)N WQDKIVRHTQYJSN-DCAQKATOSA-N 0.000 description 1
- YFQSSOAGMZGXFT-MEYUZBJRSA-N Lys-Thr-Tyr Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O YFQSSOAGMZGXFT-MEYUZBJRSA-N 0.000 description 1
- IEIHKHYMBIYQTH-YESZJQIVSA-N Lys-Tyr-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CC2=CC=C(C=C2)O)NC(=O)[C@H](CCCCN)N)C(=O)O IEIHKHYMBIYQTH-YESZJQIVSA-N 0.000 description 1
- SQRLLZAQNOQCEG-KKUMJFAQSA-N Lys-Tyr-Ser Chemical compound NCCCC[C@H](N)C(=O)N[C@H](C(=O)N[C@@H](CO)C(O)=O)CC1=CC=C(O)C=C1 SQRLLZAQNOQCEG-KKUMJFAQSA-N 0.000 description 1
- VVURYEVJJTXWNE-ULQDDVLXSA-N Lys-Tyr-Val Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](C(C)C)C(O)=O VVURYEVJJTXWNE-ULQDDVLXSA-N 0.000 description 1
- RPWQJSBMXJSCPD-XUXIUFHCSA-N Lys-Val-Ile Chemical compound CC[C@H](C)[C@H](NC(=O)[C@@H](NC(=O)[C@@H](N)CCCCN)C(C)C)C(O)=O RPWQJSBMXJSCPD-XUXIUFHCSA-N 0.000 description 1
- NYTDJEZBAAFLLG-IHRRRGAJSA-N Lys-Val-Lys Chemical compound NCCCC[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCCCN)C(O)=O NYTDJEZBAAFLLG-IHRRRGAJSA-N 0.000 description 1
- OZVXDDFYCQOPFD-XQQFMLRXSA-N Lys-Val-Pro Chemical compound CC(C)[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CCCCN)N OZVXDDFYCQOPFD-XQQFMLRXSA-N 0.000 description 1
- RIPJMCFGQHGHNP-RHYQMDGZSA-N Lys-Val-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](C(C)C)NC(=O)[C@H](CCCCN)N)O RIPJMCFGQHGHNP-RHYQMDGZSA-N 0.000 description 1
- QAHFGYLFLVGBNW-DCAQKATOSA-N Met-Ala-Lys Chemical compound CSCC[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@H](C(O)=O)CCCCN QAHFGYLFLVGBNW-DCAQKATOSA-N 0.000 description 1
- WGBMNLCRYKSWAR-DCAQKATOSA-N Met-Asp-Lys Chemical compound CSCC[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@H](C(O)=O)CCCCN WGBMNLCRYKSWAR-DCAQKATOSA-N 0.000 description 1
- FZUNSVYYPYJYAP-NAKRPEOUSA-N Met-Ile-Ala Chemical compound [H]N[C@@H](CCSC)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](C)C(O)=O FZUNSVYYPYJYAP-NAKRPEOUSA-N 0.000 description 1
- QGRJTULYDZUBAY-ZPFDUUQYSA-N Met-Ile-Glu Chemical compound [H]N[C@@H](CCSC)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CCC(O)=O)C(O)=O QGRJTULYDZUBAY-ZPFDUUQYSA-N 0.000 description 1
- AFFKUNVPPLQUGA-DCAQKATOSA-N Met-Leu-Ala Chemical compound [H]N[C@@H](CCSC)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](C)C(O)=O AFFKUNVPPLQUGA-DCAQKATOSA-N 0.000 description 1
- BEZJTLKUMFMITF-AVGNSLFASA-N Met-Lys-Arg Chemical compound CSCC[C@H](N)C(=O)N[C@@H](CCCCN)C(=O)N[C@H](C(O)=O)CCCNC(N)=N BEZJTLKUMFMITF-AVGNSLFASA-N 0.000 description 1
- KSIPKXNIQOWMIC-RCWTZXSCSA-N Met-Thr-Arg Chemical compound CSCC[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@H](C(O)=O)CCCNC(N)=N KSIPKXNIQOWMIC-RCWTZXSCSA-N 0.000 description 1
- 241001378931 Methanococcus maripaludis C7 Species 0.000 description 1
- 241000825684 Mycobacterium abscessus ATCC 19977 Species 0.000 description 1
- 241000204022 Mycoplasma gallisepticum Species 0.000 description 1
- 241000107400 Mycoplasma mobile 163K Species 0.000 description 1
- 241000051161 Mycoplasma synoviae 53 Species 0.000 description 1
- WUGMRIBZSVSJNP-UHFFFAOYSA-N N-L-alanyl-L-tryptophan Natural products C1=CC=C2C(CC(NC(=O)C(N)C)C(O)=O)=CNC2=C1 WUGMRIBZSVSJNP-UHFFFAOYSA-N 0.000 description 1
- PESQCPHRXOFIPX-UHFFFAOYSA-N N-L-methionyl-L-tyrosine Natural products CSCCC(N)C(=O)NC(C(O)=O)CC1=CC=C(O)C=C1 PESQCPHRXOFIPX-UHFFFAOYSA-N 0.000 description 1
- XMBSYZWANAQXEV-UHFFFAOYSA-N N-alpha-L-glutamyl-L-phenylalanine Natural products OC(=O)CCC(N)C(=O)NC(C(O)=O)CC1=CC=CC=C1 XMBSYZWANAQXEV-UHFFFAOYSA-N 0.000 description 1
- 108010047562 NGR peptide Proteins 0.000 description 1
- 101100068676 Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) gln-1 gene Proteins 0.000 description 1
- 241001648684 Nitrobacter hamburgensis X14 Species 0.000 description 1
- 241001037736 Nocardia farcinica IFM 10152 Species 0.000 description 1
- 102100028156 Non-homologous end-joining factor 1 Human genes 0.000 description 1
- 108010047956 Nucleosomes Proteins 0.000 description 1
- 238000002944 PCR assay Methods 0.000 description 1
- 241000601272 Parvibaculum lavamentivorans DS-1 Species 0.000 description 1
- 241000606856 Pasteurella multocida Species 0.000 description 1
- 241000549884 Persephonella marina EX-H1 Species 0.000 description 1
- CGOMLCQJEMWMCE-STQMWFEESA-N Phe-Arg-Gly Chemical compound NC(N)=NCCC[C@@H](C(=O)NCC(O)=O)NC(=O)[C@@H](N)CC1=CC=CC=C1 CGOMLCQJEMWMCE-STQMWFEESA-N 0.000 description 1
- WMGVYPPIMZPWPN-SRVKXCTJSA-N Phe-Asp-Asn Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CC(=O)O)C(=O)N[C@@H](CC(=O)N)C(=O)O)N WMGVYPPIMZPWPN-SRVKXCTJSA-N 0.000 description 1
- SWZKMTDPQXLQRD-XVSYOHENSA-N Phe-Asp-Thr Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O SWZKMTDPQXLQRD-XVSYOHENSA-N 0.000 description 1
- GDBOREPXIRKSEQ-FHWLQOOXSA-N Phe-Gln-Phe Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O GDBOREPXIRKSEQ-FHWLQOOXSA-N 0.000 description 1
- KYYMILWEGJYPQZ-IHRRRGAJSA-N Phe-Glu-Glu Chemical compound OC(=O)CC[C@@H](C(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@@H](N)CC1=CC=CC=C1 KYYMILWEGJYPQZ-IHRRRGAJSA-N 0.000 description 1
- KJJROSNFBRWPHS-JYJNAYRXSA-N Phe-Glu-Leu Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(O)=O KJJROSNFBRWPHS-JYJNAYRXSA-N 0.000 description 1
- PSKRILMFHNIUAO-JYJNAYRXSA-N Phe-Glu-Lys Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CCC(=O)O)C(=O)N[C@@H](CCCCN)C(=O)O)N PSKRILMFHNIUAO-JYJNAYRXSA-N 0.000 description 1
- WPTYDQPGBMDUBI-QWRGUYRKSA-N Phe-Gly-Asn Chemical compound N[C@@H](Cc1ccccc1)C(=O)NCC(=O)N[C@@H](CC(N)=O)C(O)=O WPTYDQPGBMDUBI-QWRGUYRKSA-N 0.000 description 1
- LRBSWBVUCLLRLU-BZSNNMDCSA-N Phe-Leu-Lys Chemical compound CC(C)C[C@H](NC(=O)[C@@H](N)Cc1ccccc1)C(=O)N[C@@H](CCCCN)C(O)=O LRBSWBVUCLLRLU-BZSNNMDCSA-N 0.000 description 1
- BSHMIVKDJQGLNT-ACRUOGEOSA-N Phe-Lys-Tyr Chemical compound C([C@H](N)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(O)=O)C1=CC=CC=C1 BSHMIVKDJQGLNT-ACRUOGEOSA-N 0.000 description 1
- TXJJXEXCZBHDNA-ACRUOGEOSA-N Phe-Phe-His Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CC2=CC=CC=C2)C(=O)N[C@@H](CC3=CN=CN3)C(=O)O)N TXJJXEXCZBHDNA-ACRUOGEOSA-N 0.000 description 1
- RBRNEFJTEHPDSL-ACRUOGEOSA-N Phe-Phe-Lys Chemical compound C([C@@H](C(=O)N[C@@H](CCCCN)C(O)=O)NC(=O)[C@@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 RBRNEFJTEHPDSL-ACRUOGEOSA-N 0.000 description 1
- DBNGDEAQXGFGRA-ACRUOGEOSA-N Phe-Tyr-Lys Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CC2=CC=C(C=C2)O)C(=O)N[C@@H](CCCCN)C(=O)O)N DBNGDEAQXGFGRA-ACRUOGEOSA-N 0.000 description 1
- YUPRIZTWANWWHK-DZKIICNBSA-N Phe-Val-Glu Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCC(=O)O)C(=O)O)NC(=O)[C@H](CC1=CC=CC=C1)N YUPRIZTWANWWHK-DZKIICNBSA-N 0.000 description 1
- OAICVXFJPJFONN-UHFFFAOYSA-N Phosphorus Chemical compound [P] OAICVXFJPJFONN-UHFFFAOYSA-N 0.000 description 1
- 229920002594 Polyethylene Glycol 8000 Polymers 0.000 description 1
- DZZCICYRSZASNF-FXQIFTODSA-N Pro-Ala-Ala Chemical compound OC(=O)[C@H](C)NC(=O)[C@H](C)NC(=O)[C@@H]1CCCN1 DZZCICYRSZASNF-FXQIFTODSA-N 0.000 description 1
- OBVCYFIHIIYIQF-CIUDSAMLSA-N Pro-Asn-Glu Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CCC(O)=O)C(O)=O OBVCYFIHIIYIQF-CIUDSAMLSA-N 0.000 description 1
- UAYHMOIGIQZLFR-NHCYSSNCSA-N Pro-Gln-Val Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](C(C)C)C(O)=O UAYHMOIGIQZLFR-NHCYSSNCSA-N 0.000 description 1
- KIPIKSXPPLABPN-CIUDSAMLSA-N Pro-Glu-Asn Chemical compound NC(=O)C[C@@H](C(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@@H]1CCCN1 KIPIKSXPPLABPN-CIUDSAMLSA-N 0.000 description 1
- MGDFPGCFVJFITQ-CIUDSAMLSA-N Pro-Glu-Asp Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(O)=O)C(O)=O MGDFPGCFVJFITQ-CIUDSAMLSA-N 0.000 description 1
- VDGTVWFMRXVQCT-GUBZILKMSA-N Pro-Glu-Gln Chemical compound NC(=O)CC[C@@H](C(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@@H]1CCCN1 VDGTVWFMRXVQCT-GUBZILKMSA-N 0.000 description 1
- VOZIBWWZSBIXQN-SRVKXCTJSA-N Pro-Glu-Lys Chemical compound NCCCC[C@H](NC(=O)[C@H](CCC(O)=O)NC(=O)[C@@H]1CCCN1)C(O)=O VOZIBWWZSBIXQN-SRVKXCTJSA-N 0.000 description 1
- VZKBJNBZMZHKRC-XUXIUFHCSA-N Pro-Ile-Leu Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC(C)C)C(O)=O VZKBJNBZMZHKRC-XUXIUFHCSA-N 0.000 description 1
- CDGABSWLRMECHC-IHRRRGAJSA-N Pro-Lys-His Chemical compound C1C[C@H](NC1)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC2=CN=CN2)C(=O)O CDGABSWLRMECHC-IHRRRGAJSA-N 0.000 description 1
- FNGOXVQBBCMFKV-CIUDSAMLSA-N Pro-Ser-Glu Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CO)C(=O)N[C@@H](CCC(O)=O)C(O)=O FNGOXVQBBCMFKV-CIUDSAMLSA-N 0.000 description 1
- QDDJNKWPTJHROJ-UFYCRDLUSA-N Pro-Tyr-Tyr Chemical compound C([C@@H](C(=O)O)NC(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)[C@H]1NCCC1)C1=CC=C(O)C=C1 QDDJNKWPTJHROJ-UFYCRDLUSA-N 0.000 description 1
- 241000773205 Pseudarthrobacter chlorophenolicus A6 Species 0.000 description 1
- 241000695265 Pseudoalteromonas atlantica T6c Species 0.000 description 1
- 108010003201 RGH 0205 Proteins 0.000 description 1
- 102000044126 RNA-Binding Proteins Human genes 0.000 description 1
- 108700020471 RNA-Binding Proteins Proteins 0.000 description 1
- 241000647111 Rhodococcus erythropolis PR4 Species 0.000 description 1
- 241001459443 Rhodococcus jostii RHA1 Species 0.000 description 1
- 241001113889 Rhodococcus opacus B4 Species 0.000 description 1
- 241001303431 Rhodopseudomonas palustris BisB5 Species 0.000 description 1
- 241000134686 Rhodospirillum rubrum ATCC 11170 Species 0.000 description 1
- 102000003661 Ribonuclease III Human genes 0.000 description 1
- 108010057163 Ribonuclease III Proteins 0.000 description 1
- 102000004389 Ribonucleoproteins Human genes 0.000 description 1
- 108010081734 Ribonucleoproteins Proteins 0.000 description 1
- 241000516659 Roseiflexus Species 0.000 description 1
- 241000504328 Roseiflexus castenholzii DSM 13941 Species 0.000 description 1
- 239000006146 Roswell Park Memorial Institute medium Substances 0.000 description 1
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 1
- BTKUIVBNGBFTTP-WHFBIAKZSA-N Ser-Ala-Gly Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](C)C(=O)NCC(O)=O BTKUIVBNGBFTTP-WHFBIAKZSA-N 0.000 description 1
- QFBNNYNWKYKVJO-DCAQKATOSA-N Ser-Arg-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)[C@@H](N)CO)CCCN=C(N)N QFBNNYNWKYKVJO-DCAQKATOSA-N 0.000 description 1
- ZXLUWXWISXIFIX-ACZMJKKPSA-N Ser-Asn-Glu Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CCC(O)=O)C(O)=O ZXLUWXWISXIFIX-ACZMJKKPSA-N 0.000 description 1
- BNFVPSRLHHPQKS-WHFBIAKZSA-N Ser-Asp-Gly Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(O)=O)C(=O)NCC(O)=O BNFVPSRLHHPQKS-WHFBIAKZSA-N 0.000 description 1
- OLIJLNWFEQEFDM-SRVKXCTJSA-N Ser-Asp-Phe Chemical compound OC[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 OLIJLNWFEQEFDM-SRVKXCTJSA-N 0.000 description 1
- OJPHFSOMBZKQKQ-GUBZILKMSA-N Ser-Gln-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CCC(N)=O)NC(=O)[C@@H](N)CO OJPHFSOMBZKQKQ-GUBZILKMSA-N 0.000 description 1
- LALNXSXEYFUUDD-GUBZILKMSA-N Ser-Glu-Leu Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(C)C)C(O)=O LALNXSXEYFUUDD-GUBZILKMSA-N 0.000 description 1
- DSGYZICNAMEJOC-AVGNSLFASA-N Ser-Glu-Phe Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O DSGYZICNAMEJOC-AVGNSLFASA-N 0.000 description 1
- SNVIOQXAHVORQM-WDSKDSINSA-N Ser-Gly-Gln Chemical compound [H]N[C@@H](CO)C(=O)NCC(=O)N[C@@H](CCC(N)=O)C(O)=O SNVIOQXAHVORQM-WDSKDSINSA-N 0.000 description 1
- WEQAYODCJHZSJZ-KKUMJFAQSA-N Ser-His-Tyr Chemical compound C([C@H](NC(=O)[C@H](CO)N)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(O)=O)C1=CN=CN1 WEQAYODCJHZSJZ-KKUMJFAQSA-N 0.000 description 1
- JIPVNVNKXJLFJF-BJDJZHNGSA-N Ser-Ile-Lys Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CO)N JIPVNVNKXJLFJF-BJDJZHNGSA-N 0.000 description 1
- ZOPISOXXPQNOCO-SVSWQMSJSA-N Ser-Ile-Thr Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H]([C@@H](C)O)C(=O)O)NC(=O)[C@H](CO)N ZOPISOXXPQNOCO-SVSWQMSJSA-N 0.000 description 1
- MUJQWSAWLLRJCE-KATARQTJSA-N Ser-Leu-Thr Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O MUJQWSAWLLRJCE-KATARQTJSA-N 0.000 description 1
- CRJZZXMAADSBBQ-SRVKXCTJSA-N Ser-Lys-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@H](CCCCN)NC(=O)[C@@H](N)CO CRJZZXMAADSBBQ-SRVKXCTJSA-N 0.000 description 1
- UPLYXVPQLJVWMM-KKUMJFAQSA-N Ser-Phe-Leu Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CC(C)C)C(O)=O UPLYXVPQLJVWMM-KKUMJFAQSA-N 0.000 description 1
- ADJDNJCSPNFFPI-FXQIFTODSA-N Ser-Pro-Ala Chemical compound OC(=O)[C@H](C)NC(=O)[C@@H]1CCCN1C(=O)[C@@H](N)CO ADJDNJCSPNFFPI-FXQIFTODSA-N 0.000 description 1
- FLONGDPORFIVQW-XGEHTFHBSA-N Ser-Pro-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)[C@@H]1CCCN1C(=O)[C@@H](N)CO FLONGDPORFIVQW-XGEHTFHBSA-N 0.000 description 1
- WUXCHQZLUHBSDJ-LKXGYXEUSA-N Ser-Thr-Asp Chemical compound OC[C@H](N)C(=O)N[C@@H]([C@H](O)C)C(=O)N[C@@H](CC(O)=O)C(O)=O WUXCHQZLUHBSDJ-LKXGYXEUSA-N 0.000 description 1
- BEBVVQPDSHHWQL-NRPADANISA-N Ser-Val-Glu Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(O)=O)C(O)=O BEBVVQPDSHHWQL-NRPADANISA-N 0.000 description 1
- LGIMRDKGABDMBN-DCAQKATOSA-N Ser-Val-Lys Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CO)N LGIMRDKGABDMBN-DCAQKATOSA-N 0.000 description 1
- 241000933177 Shewanella pealeana ATCC 700345 Species 0.000 description 1
- 241001496704 Slackia heliotrinireducens DSM 20476 Species 0.000 description 1
- 241000756832 Streptobacillus moniliformis DSM 12112 Species 0.000 description 1
- 241000193985 Streptococcus agalactiae Species 0.000 description 1
- 241001209210 Streptococcus agalactiae A909 Species 0.000 description 1
- 241001540742 Streptococcus agalactiae NEM316 Species 0.000 description 1
- 241000194042 Streptococcus dysgalactiae Species 0.000 description 1
- 241000120569 Streptococcus equi subsp. zooepidemicus Species 0.000 description 1
- 241001167808 Streptococcus gallolyticus UCN34 Species 0.000 description 1
- 241000194026 Streptococcus gordonii Species 0.000 description 1
- 241001147754 Streptococcus gordonii str. Challis Species 0.000 description 1
- 241000194019 Streptococcus mutans Species 0.000 description 1
- 241000672607 Streptococcus mutans NN2025 Species 0.000 description 1
- 241000320123 Streptococcus pyogenes M1 GAS Species 0.000 description 1
- 241000103155 Streptococcus pyogenes MGAS10270 Species 0.000 description 1
- 241000103160 Streptococcus pyogenes MGAS10750 Species 0.000 description 1
- 241000103154 Streptococcus pyogenes MGAS2096 Species 0.000 description 1
- 241001520169 Streptococcus pyogenes MGAS315 Species 0.000 description 1
- 241001148739 Streptococcus pyogenes MGAS5005 Species 0.000 description 1
- 241001332083 Streptococcus pyogenes MGAS6180 Species 0.000 description 1
- 241000103156 Streptococcus pyogenes MGAS9429 Species 0.000 description 1
- 241001496716 Streptococcus pyogenes NZ131 Species 0.000 description 1
- 241001455236 Streptococcus pyogenes SSI-1 Species 0.000 description 1
- 241000192593 Synechocystis sp. PCC 6803 Species 0.000 description 1
- 108010006785 Taq Polymerase Proteins 0.000 description 1
- 241001496699 Thermomonospora curvata DSM 43183 Species 0.000 description 1
- FQPQPTHMHZKGFM-XQXXSGGOSA-N Thr-Ala-Glu Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C)C(=O)N[C@@H](CCC(O)=O)C(O)=O FQPQPTHMHZKGFM-XQXXSGGOSA-N 0.000 description 1
- ZUXQFMVPAYGPFJ-JXUBOQSCSA-N Thr-Ala-Lys Chemical compound C[C@@H](O)[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@H](C(O)=O)CCCCN ZUXQFMVPAYGPFJ-JXUBOQSCSA-N 0.000 description 1
- CEXFELBFVHLYDZ-XGEHTFHBSA-N Thr-Arg-Ser Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CO)C(O)=O CEXFELBFVHLYDZ-XGEHTFHBSA-N 0.000 description 1
- IRKWVRSEQFTGGV-VEVYYDQMSA-N Thr-Asn-Arg Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O IRKWVRSEQFTGGV-VEVYYDQMSA-N 0.000 description 1
- OJRNZRROAIAHDL-LKXGYXEUSA-N Thr-Asn-Ser Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CO)C(O)=O OJRNZRROAIAHDL-LKXGYXEUSA-N 0.000 description 1
- QILPDQCTQZDHFM-HJGDQZAQSA-N Thr-Gln-Arg Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O QILPDQCTQZDHFM-HJGDQZAQSA-N 0.000 description 1
- RKDFEMGVMMYYNG-WDCWCFNPSA-N Thr-Gln-Leu Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(C)C)C(O)=O RKDFEMGVMMYYNG-WDCWCFNPSA-N 0.000 description 1
- JMGJDTNUMAZNLX-RWRJDSDZSA-N Thr-Glu-Ile Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O JMGJDTNUMAZNLX-RWRJDSDZSA-N 0.000 description 1
- AQAMPXBRJJWPNI-JHEQGTHGSA-N Thr-Gly-Glu Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)NCC(=O)N[C@@H](CCC(O)=O)C(O)=O AQAMPXBRJJWPNI-JHEQGTHGSA-N 0.000 description 1
- XPNSAQMEAVSQRD-FBCQKBJTSA-N Thr-Gly-Gly Chemical compound C[C@@H](O)[C@H](N)C(=O)NCC(=O)NCC(O)=O XPNSAQMEAVSQRD-FBCQKBJTSA-N 0.000 description 1
- YSXYEJWDHBCTDJ-DVJZZOLTSA-N Thr-Gly-Trp Chemical compound C[C@H]([C@@H](C(=O)NCC(=O)N[C@@H](CC1=CNC2=CC=CC=C21)C(=O)O)N)O YSXYEJWDHBCTDJ-DVJZZOLTSA-N 0.000 description 1
- BVOVIGCHYNFJBZ-JXUBOQSCSA-N Thr-Leu-Ala Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](C)C(O)=O BVOVIGCHYNFJBZ-JXUBOQSCSA-N 0.000 description 1
- MECLEFZMPPOEAC-VOAKCMCISA-N Thr-Leu-Lys Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(=O)O)N)O MECLEFZMPPOEAC-VOAKCMCISA-N 0.000 description 1
- SCSVNSNWUTYSFO-WDCWCFNPSA-N Thr-Lys-Glu Chemical compound C[C@@H](O)[C@H](N)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCC(O)=O)C(O)=O SCSVNSNWUTYSFO-WDCWCFNPSA-N 0.000 description 1
- KKPOGALELPLJTL-MEYUZBJRSA-N Thr-Lys-Tyr Chemical compound C[C@@H](O)[C@H](N)C(=O)N[C@@H](CCCCN)C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 KKPOGALELPLJTL-MEYUZBJRSA-N 0.000 description 1
- WRQLCVIALDUQEQ-UNQGMJICSA-N Thr-Phe-Arg Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O WRQLCVIALDUQEQ-UNQGMJICSA-N 0.000 description 1
- WYLAVUAWOUVUCA-XVSYOHENSA-N Thr-Phe-Asp Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CC(O)=O)C(O)=O WYLAVUAWOUVUCA-XVSYOHENSA-N 0.000 description 1
- IWAVRIPRTCJAQO-HSHDSVGOSA-N Thr-Pro-Trp Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(O)=O IWAVRIPRTCJAQO-HSHDSVGOSA-N 0.000 description 1
- MFMGPEKYBXFIRF-SUSMZKCASA-N Thr-Thr-Gln Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCC(N)=O)C(O)=O MFMGPEKYBXFIRF-SUSMZKCASA-N 0.000 description 1
- NHQVWACSJZJCGJ-FLBSBUHZSA-N Thr-Thr-Ile Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O NHQVWACSJZJCGJ-FLBSBUHZSA-N 0.000 description 1
- ABCLYRRGTZNIFU-BWAGICSOSA-N Thr-Tyr-His Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)N[C@@H](CC2=CN=CN2)C(=O)O)N)O ABCLYRRGTZNIFU-BWAGICSOSA-N 0.000 description 1
- PWONLXBUSVIZPH-RHYQMDGZSA-N Thr-Val-Lys Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCCCN)C(=O)O)N)O PWONLXBUSVIZPH-RHYQMDGZSA-N 0.000 description 1
- 108091028113 Trans-activating crRNA Proteins 0.000 description 1
- 241000999858 Treponema denticola ATCC 35405 Species 0.000 description 1
- 239000007983 Tris buffer Substances 0.000 description 1
- DLZKEQQWXODGGZ-KWQFWETISA-N Tyr-Ala-Gly Chemical compound OC(=O)CNC(=O)[C@H](C)NC(=O)[C@@H](N)CC1=CC=C(O)C=C1 DLZKEQQWXODGGZ-KWQFWETISA-N 0.000 description 1
- AYPAIRCDLARHLM-KKUMJFAQSA-N Tyr-Asn-Lys Chemical compound C1=CC(=CC=C1C[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H](CCCCN)C(=O)O)N)O AYPAIRCDLARHLM-KKUMJFAQSA-N 0.000 description 1
- UABYBEBXFFNCIR-YDHLFZDLSA-N Tyr-Asp-Val Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](C(C)C)C(O)=O UABYBEBXFFNCIR-YDHLFZDLSA-N 0.000 description 1
- KIJLSRYAUGGZIN-CFMVVWHZSA-N Tyr-Ile-Asp Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC(O)=O)C(O)=O KIJLSRYAUGGZIN-CFMVVWHZSA-N 0.000 description 1
- NKUGCYDFQKFVOJ-JYJNAYRXSA-N Tyr-Leu-Gln Chemical compound NC(=O)CC[C@@H](C(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@@H](N)CC1=CC=C(O)C=C1 NKUGCYDFQKFVOJ-JYJNAYRXSA-N 0.000 description 1
- JLKVWTICWVWGSK-JYJNAYRXSA-N Tyr-Lys-Glu Chemical compound OC(=O)CC[C@@H](C(O)=O)NC(=O)[C@H](CCCCN)NC(=O)[C@@H](N)CC1=CC=C(O)C=C1 JLKVWTICWVWGSK-JYJNAYRXSA-N 0.000 description 1
- PMHLLBKTDHQMCY-ULQDDVLXSA-N Tyr-Lys-Val Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](C(C)C)C(O)=O PMHLLBKTDHQMCY-ULQDDVLXSA-N 0.000 description 1
- JXGUUJMPCRXMSO-HJOGWXRNSA-N Tyr-Phe-Phe Chemical compound C([C@H](N)C(=O)N[C@@H](CC=1C=CC=CC=1)C(=O)N[C@@H](CC=1C=CC=CC=1)C(O)=O)C1=CC=C(O)C=C1 JXGUUJMPCRXMSO-HJOGWXRNSA-N 0.000 description 1
- VBFVQTPETKJCQW-RPTUDFQQSA-N Tyr-Phe-Thr Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H]([C@@H](C)O)C(O)=O VBFVQTPETKJCQW-RPTUDFQQSA-N 0.000 description 1
- RCMWNNJFKNDKQR-UFYCRDLUSA-N Tyr-Pro-Phe Chemical compound C([C@H](N)C(=O)N1[C@@H](CCC1)C(=O)N[C@@H](CC=1C=CC=CC=1)C(O)=O)C1=CC=C(O)C=C1 RCMWNNJFKNDKQR-UFYCRDLUSA-N 0.000 description 1
- XGZBEGGGAUQBMB-KJEVXHAQSA-N Tyr-Pro-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@@H]1CCCN1C(=O)[C@H](CC2=CC=C(C=C2)O)N)O XGZBEGGGAUQBMB-KJEVXHAQSA-N 0.000 description 1
- SOAUMCDLIUGXJJ-SRVKXCTJSA-N Tyr-Ser-Asn Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(N)=O)C(O)=O SOAUMCDLIUGXJJ-SRVKXCTJSA-N 0.000 description 1
- MQGGXGKQSVEQHR-KKUMJFAQSA-N Tyr-Ser-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CC1=CC=C(O)C=C1 MQGGXGKQSVEQHR-KKUMJFAQSA-N 0.000 description 1
- SQUMHUZLJDUROQ-YDHLFZDLSA-N Tyr-Val-Asp Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CC(O)=O)C(O)=O SQUMHUZLJDUROQ-YDHLFZDLSA-N 0.000 description 1
- ABSXSJZNRAQDDI-KJEVXHAQSA-N Tyr-Val-Thr Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O ABSXSJZNRAQDDI-KJEVXHAQSA-N 0.000 description 1
- 101100316831 Vaccinia virus (strain Copenhagen) B18R gene Proteins 0.000 description 1
- 101100004099 Vaccinia virus (strain Western Reserve) VACWR200 gene Proteins 0.000 description 1
- RUCNAYOMFXRIKJ-DCAQKATOSA-N Val-Ala-Lys Chemical compound CC(C)[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@H](C(O)=O)CCCCN RUCNAYOMFXRIKJ-DCAQKATOSA-N 0.000 description 1
- PAPWZOJOLKZEFR-AVGNSLFASA-N Val-Arg-Lys Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CCCCN)C(=O)O)N PAPWZOJOLKZEFR-AVGNSLFASA-N 0.000 description 1
- PVPAOIGJYHVWBT-KKHAAJSZSA-N Val-Asn-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CC(=O)N)NC(=O)[C@H](C(C)C)N)O PVPAOIGJYHVWBT-KKHAAJSZSA-N 0.000 description 1
- HZYOWMGWKKRMBZ-BYULHYEWSA-N Val-Asp-Asp Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC(=O)O)C(=O)N[C@@H](CC(=O)O)C(=O)O)N HZYOWMGWKKRMBZ-BYULHYEWSA-N 0.000 description 1
- VLOYGOZDPGYWFO-LAEOZQHASA-N Val-Asp-Glu Chemical compound CC(C)[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CCC(O)=O)C(O)=O VLOYGOZDPGYWFO-LAEOZQHASA-N 0.000 description 1
- BMGOFDMKDVVGJG-NHCYSSNCSA-N Val-Asp-Lys Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC(=O)O)C(=O)N[C@@H](CCCCN)C(=O)O)N BMGOFDMKDVVGJG-NHCYSSNCSA-N 0.000 description 1
- XTAUQCGQFJQGEJ-NHCYSSNCSA-N Val-Gln-Arg Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)N[C@@H](CCCN=C(N)N)C(=O)O)N XTAUQCGQFJQGEJ-NHCYSSNCSA-N 0.000 description 1
- NYTKXWLZSNRILS-IFFSRLJSSA-N Val-Gln-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](C(C)C)N)O NYTKXWLZSNRILS-IFFSRLJSSA-N 0.000 description 1
- CVIXTAITYJQMPE-LAEOZQHASA-N Val-Glu-Asn Chemical compound CC(C)[C@H](N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(N)=O)C(O)=O CVIXTAITYJQMPE-LAEOZQHASA-N 0.000 description 1
- GBESYURLQOYWLU-LAEOZQHASA-N Val-Glu-Asp Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCC(=O)O)C(=O)N[C@@H](CC(=O)O)C(=O)O)N GBESYURLQOYWLU-LAEOZQHASA-N 0.000 description 1
- ZXAGTABZUOMUDO-GVXVVHGQSA-N Val-Glu-Lys Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCC(=O)O)C(=O)N[C@@H](CCCCN)C(=O)O)N ZXAGTABZUOMUDO-GVXVVHGQSA-N 0.000 description 1
- XWYUBUYQMOUFRQ-IFFSRLJSSA-N Val-Glu-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)O)NC(=O)[C@H](C(C)C)N)O XWYUBUYQMOUFRQ-IFFSRLJSSA-N 0.000 description 1
- YTPLVNUZZOBFFC-SCZZXKLOSA-N Val-Gly-Pro Chemical compound CC(C)[C@H](N)C(=O)NCC(=O)N1CCC[C@@H]1C(O)=O YTPLVNUZZOBFFC-SCZZXKLOSA-N 0.000 description 1
- KZKMBGXCNLPYKD-YEPSODPASA-N Val-Gly-Thr Chemical compound CC(C)[C@H](N)C(=O)NCC(=O)N[C@@H]([C@@H](C)O)C(O)=O KZKMBGXCNLPYKD-YEPSODPASA-N 0.000 description 1
- APQIVBCUIUDSMB-OSUNSFLBSA-N Val-Ile-Thr Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H]([C@@H](C)O)C(=O)O)NC(=O)[C@H](C(C)C)N APQIVBCUIUDSMB-OSUNSFLBSA-N 0.000 description 1
- AGXGCFSECFQMKB-NHCYSSNCSA-N Val-Leu-Asp Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC(=O)O)C(=O)O)NC(=O)[C@H](C(C)C)N AGXGCFSECFQMKB-NHCYSSNCSA-N 0.000 description 1
- WBAJDGWKRIHOAC-GVXVVHGQSA-N Val-Lys-Gln Chemical compound [H]N[C@@H](C(C)C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCC(N)=O)C(O)=O WBAJDGWKRIHOAC-GVXVVHGQSA-N 0.000 description 1
- ZRSZTKTVPNSUNA-IHRRRGAJSA-N Val-Lys-Leu Chemical compound CC(C)C[C@H](NC(=O)[C@H](CCCCN)NC(=O)[C@@H](N)C(C)C)C(O)=O ZRSZTKTVPNSUNA-IHRRRGAJSA-N 0.000 description 1
- JVGHIFMSFBZDHH-WPRPVWTQSA-N Val-Met-Gly Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCSC)C(=O)NCC(=O)O)N JVGHIFMSFBZDHH-WPRPVWTQSA-N 0.000 description 1
- UEPLNXPLHJUYPT-AVGNSLFASA-N Val-Met-Lys Chemical compound CC(C)[C@H](N)C(=O)N[C@@H](CCSC)C(=O)N[C@@H](CCCCN)C(O)=O UEPLNXPLHJUYPT-AVGNSLFASA-N 0.000 description 1
- GQMNEJMFMCJJTD-NHCYSSNCSA-N Val-Pro-Gln Chemical compound CC(C)[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CCC(N)=O)C(O)=O GQMNEJMFMCJJTD-NHCYSSNCSA-N 0.000 description 1
- MIAZWUMFUURQNP-YDHLFZDLSA-N Val-Tyr-Asn Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)N[C@@H](CC(=O)N)C(=O)O)N MIAZWUMFUURQNP-YDHLFZDLSA-N 0.000 description 1
- VTIAEOKFUJJBTC-YDHLFZDLSA-N Val-Tyr-Asp Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)N[C@@H](CC(=O)O)C(=O)O)N VTIAEOKFUJJBTC-YDHLFZDLSA-N 0.000 description 1
- GUIYPEKUEMQBIK-JSGCOSHPSA-N Val-Tyr-Gly Chemical compound CC(C)[C@H](N)C(=O)N[C@@H](Cc1ccc(O)cc1)C(=O)NCC(O)=O GUIYPEKUEMQBIK-JSGCOSHPSA-N 0.000 description 1
- AOILQMZPNLUXCM-AVGNSLFASA-N Val-Val-Lys Chemical compound CC(C)[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@H](C(O)=O)CCCCN AOILQMZPNLUXCM-AVGNSLFASA-N 0.000 description 1
- 241000847071 Verminephrobacter eiseniae EF01-2 Species 0.000 description 1
- 108700005077 Viral Genes Proteins 0.000 description 1
- 241000605939 Wolinella succinogenes Species 0.000 description 1
- 241000883281 [Clostridium] cellulolyticum H10 Species 0.000 description 1
- 241000714896 [Eubacterium] rectale ATCC 33656 Species 0.000 description 1
- 108010076089 accutase Proteins 0.000 description 1
- 108010081404 acein-2 Proteins 0.000 description 1
- 239000008186 active pharmaceutical agent Substances 0.000 description 1
- 230000004721 adaptive immunity Effects 0.000 description 1
- 101150063416 add gene Proteins 0.000 description 1
- 239000011543 agarose gel Substances 0.000 description 1
- 238000000246 agarose gel electrophoresis Methods 0.000 description 1
- 108010008685 alanyl-glutamyl-aspartic acid Proteins 0.000 description 1
- 108010024078 alanyl-glycyl-serine Proteins 0.000 description 1
- 108010086434 alanyl-seryl-glycine Proteins 0.000 description 1
- 108010087924 alanylproline Proteins 0.000 description 1
- 208000026935 allergic disease Diseases 0.000 description 1
- 125000000539 amino acid group Chemical group 0.000 description 1
- 230000003321 amplification Effects 0.000 description 1
- 239000003242 anti bacterial agent Substances 0.000 description 1
- 229940088710 antibiotic agent Drugs 0.000 description 1
- 108010008355 arginyl-glutamine Proteins 0.000 description 1
- 108010001271 arginyl-glutamyl-arginine Proteins 0.000 description 1
- 108010059459 arginyl-threonyl-phenylalanine Proteins 0.000 description 1
- 108010084758 arginyl-tyrosyl-aspartic acid Proteins 0.000 description 1
- 238000003491 array Methods 0.000 description 1
- 108010069205 aspartyl-phenylalanine Proteins 0.000 description 1
- 108010093581 aspartyl-proline Proteins 0.000 description 1
- 108010038633 aspartylglutamate Proteins 0.000 description 1
- 108010047857 aspartylglycine Proteins 0.000 description 1
- 108010068265 aspartyltyrosine Proteins 0.000 description 1
- 230000001580 bacterial effect Effects 0.000 description 1
- 230000004888 barrier function Effects 0.000 description 1
- 230000009286 beneficial effect Effects 0.000 description 1
- 238000000876 binomial test Methods 0.000 description 1
- 230000033228 biological regulation Effects 0.000 description 1
- 230000015556 catabolic process Effects 0.000 description 1
- 108020001778 catalytic domains Proteins 0.000 description 1
- 101150102092 ccdB gene Proteins 0.000 description 1
- 238000004113 cell culture Methods 0.000 description 1
- 210000002421 cell wall Anatomy 0.000 description 1
- 230000001413 cellular effect Effects 0.000 description 1
- 239000003153 chemical reaction reagent Substances 0.000 description 1
- 239000013611 chromosomal DNA Substances 0.000 description 1
- 230000002759 chromosomal effect Effects 0.000 description 1
- 230000008711 chromosomal rearrangement Effects 0.000 description 1
- 238000012411 cloning technique Methods 0.000 description 1
- 239000013599 cloning vector Substances 0.000 description 1
- 229960002424 collagenase Drugs 0.000 description 1
- 210000001072 colon Anatomy 0.000 description 1
- 230000005757 colony formation Effects 0.000 description 1
- 238000012790 confirmation Methods 0.000 description 1
- 230000021615 conjugation Effects 0.000 description 1
- 238000010219 correlation analysis Methods 0.000 description 1
- 230000008878 coupling Effects 0.000 description 1
- 238000010168 coupling process Methods 0.000 description 1
- 238000005859 coupling reaction Methods 0.000 description 1
- 238000012258 culturing Methods 0.000 description 1
- 238000005520 cutting process Methods 0.000 description 1
- 231100000135 cytotoxicity Toxicity 0.000 description 1
- 230000003013 cytotoxicity Effects 0.000 description 1
- 238000007405 data analysis Methods 0.000 description 1
- 230000007123 defense Effects 0.000 description 1
- 230000007812 deficiency Effects 0.000 description 1
- 230000002950 deficient Effects 0.000 description 1
- 238000006731 degradation reaction Methods 0.000 description 1
- 230000000593 degrading effect Effects 0.000 description 1
- 238000010586 diagram Methods 0.000 description 1
- 230000004069 differentiation Effects 0.000 description 1
- 201000010099 disease Diseases 0.000 description 1
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 1
- ZGSPNIOCEDOHGS-UHFFFAOYSA-L disodium [3-[2,3-di(octadeca-9,12-dienoyloxy)propoxy-oxidophosphoryl]oxy-2-hydroxypropyl] 2,3-di(octadeca-9,12-dienoyloxy)propyl phosphate Chemical compound [Na+].[Na+].CCCCCC=CCC=CCCCCCCCC(=O)OCC(OC(=O)CCCCCCCC=CCC=CCCCCC)COP([O-])(=O)OCC(O)COP([O-])(=O)OCC(OC(=O)CCCCCCCC=CCC=CCCCCC)COC(=O)CCCCCCCC=CCC=CCCCCC ZGSPNIOCEDOHGS-UHFFFAOYSA-L 0.000 description 1
- 229940079593 drug Drugs 0.000 description 1
- 239000003814 drug Substances 0.000 description 1
- 238000007877 drug screening Methods 0.000 description 1
- 210000001671 embryonic stem cell Anatomy 0.000 description 1
- 108010026638 endodeoxyribonuclease FokI Proteins 0.000 description 1
- 238000005516 engineering process Methods 0.000 description 1
- 230000004049 epigenetic modification Effects 0.000 description 1
- 230000003203 everyday effect Effects 0.000 description 1
- 230000001747 exhibiting effect Effects 0.000 description 1
- 108010052305 exodeoxyribonuclease III Proteins 0.000 description 1
- 238000000605 extraction Methods 0.000 description 1
- 210000002950 fibroblast Anatomy 0.000 description 1
- 238000001914 filtration Methods 0.000 description 1
- 231100000221 frame shift mutation induction Toxicity 0.000 description 1
- 230000037433 frameshift Effects 0.000 description 1
- 230000002538 fungal effect Effects 0.000 description 1
- 238000003209 gene knockout Methods 0.000 description 1
- 238000012239 gene modification Methods 0.000 description 1
- 238000010363 gene targeting Methods 0.000 description 1
- 108091006104 gene-regulatory proteins Proteins 0.000 description 1
- 102000034356 gene-regulatory proteins Human genes 0.000 description 1
- 230000005017 genetic modification Effects 0.000 description 1
- 235000013617 genetically modified food Nutrition 0.000 description 1
- 230000037442 genomic alteration Effects 0.000 description 1
- 210000001654 germ layer Anatomy 0.000 description 1
- 239000008103 glucose Substances 0.000 description 1
- 108010079547 glutamylmethionine Proteins 0.000 description 1
- HPAIKDPJURGQLN-UHFFFAOYSA-N glycyl-L-histidyl-L-phenylalanine Natural products C=1C=CC=CC=1CC(C(O)=O)NC(=O)C(NC(=O)CN)CC1=CN=CN1 HPAIKDPJURGQLN-UHFFFAOYSA-N 0.000 description 1
- XBGGUPMXALFZOT-UHFFFAOYSA-N glycyl-L-tyrosine hemihydrate Natural products NCC(=O)NC(C(O)=O)CC1=CC=C(O)C=C1 XBGGUPMXALFZOT-UHFFFAOYSA-N 0.000 description 1
- 108010000434 glycyl-alanyl-leucine Proteins 0.000 description 1
- 108010026364 glycyl-glycyl-leucine Proteins 0.000 description 1
- 108010050475 glycyl-leucyl-tyrosine Proteins 0.000 description 1
- 108010089804 glycyl-threonine Proteins 0.000 description 1
- 108010048994 glycyl-tyrosyl-alanine Proteins 0.000 description 1
- 108010081551 glycylphenylalanine Proteins 0.000 description 1
- 108010087823 glycyltyrosine Proteins 0.000 description 1
- 108010037850 glycylvaline Proteins 0.000 description 1
- 239000000833 heterodimer Substances 0.000 description 1
- 108010036413 histidylglycine Proteins 0.000 description 1
- 108010028295 histidylhistidine Proteins 0.000 description 1
- 108010085325 histidylproline Proteins 0.000 description 1
- 230000009610 hypersensitivity Effects 0.000 description 1
- 230000036039 immunity Effects 0.000 description 1
- 238000003125 immunofluorescent labeling Methods 0.000 description 1
- 230000006872 improvement Effects 0.000 description 1
- 230000005764 inhibitory process Effects 0.000 description 1
- 238000002347 injection Methods 0.000 description 1
- 239000007924 injection Substances 0.000 description 1
- 238000002955 isolation Methods 0.000 description 1
- 108010027338 isoleucylcysteine Proteins 0.000 description 1
- 238000005304 joining Methods 0.000 description 1
- 229940017800 lactobacillus casei Drugs 0.000 description 1
- 229940059406 lactobacillus rhamnosus gg Drugs 0.000 description 1
- 108010044311 leucyl-glycyl-glycine Proteins 0.000 description 1
- 108010030617 leucyl-phenylalanyl-valine Proteins 0.000 description 1
- 108010000761 leucylarginine Proteins 0.000 description 1
- 108010012058 leucyltyrosine Proteins 0.000 description 1
- 238000001638 lipofection Methods 0.000 description 1
- 239000002502 liposome Substances 0.000 description 1
- 230000004807 localization Effects 0.000 description 1
- 210000003141 lower extremity Anatomy 0.000 description 1
- 239000012139 lysis buffer Substances 0.000 description 1
- 108010003700 lysyl aspartic acid Proteins 0.000 description 1
- 108010044348 lysyl-glutamyl-aspartic acid Proteins 0.000 description 1
- 108010045397 lysyl-tyrosyl-lysine Proteins 0.000 description 1
- 108010009298 lysylglutamic acid Proteins 0.000 description 1
- 108010038320 lysylphenylalanine Proteins 0.000 description 1
- 229910001629 magnesium chloride Inorganic materials 0.000 description 1
- 238000012423 maintenance Methods 0.000 description 1
- 210000004962 mammalian cell Anatomy 0.000 description 1
- 238000004519 manufacturing process Methods 0.000 description 1
- 238000013507 mapping Methods 0.000 description 1
- 230000035800 maturation Effects 0.000 description 1
- 201000000271 mature teratoma Diseases 0.000 description 1
- 238000002844 melting Methods 0.000 description 1
- 230000008018 melting Effects 0.000 description 1
- 238000000520 microinjection Methods 0.000 description 1
- 231100000324 minimal toxicity Toxicity 0.000 description 1
- 210000003470 mitochondria Anatomy 0.000 description 1
- -1 mitochondria Chemical compound 0.000 description 1
- DOBKQCZBPPCLEG-UHFFFAOYSA-N n-benzyl-2-(pyrimidin-4-ylamino)-1,3-thiazole-4-carboxamide Chemical compound C=1SC(NC=2N=CN=CC=2)=NC=1C(=O)NCC1=CC=CC=C1 DOBKQCZBPPCLEG-UHFFFAOYSA-N 0.000 description 1
- 239000002105 nanoparticle Substances 0.000 description 1
- 238000010899 nucleation Methods 0.000 description 1
- 238000003199 nucleic acid amplification method Methods 0.000 description 1
- 210000001623 nucleosome Anatomy 0.000 description 1
- 210000004940 nucleus Anatomy 0.000 description 1
- 229940124276 oligodeoxyribonucleotide Drugs 0.000 description 1
- 238000005580 one pot reaction Methods 0.000 description 1
- 238000004806 packaging method and process Methods 0.000 description 1
- 239000002245 particle Substances 0.000 description 1
- 229940051027 pasteurella multocida Drugs 0.000 description 1
- 108010064486 phenylalanyl-leucyl-valine Proteins 0.000 description 1
- 108010051242 phenylalanylserine Proteins 0.000 description 1
- 229910052698 phosphorus Inorganic materials 0.000 description 1
- 239000011574 phosphorus Substances 0.000 description 1
- 108010025488 pinealon Proteins 0.000 description 1
- 229910052697 platinum Inorganic materials 0.000 description 1
- 210000001236 prokaryotic cell Anatomy 0.000 description 1
- 108010031719 prolyl-serine Proteins 0.000 description 1
- 108010070643 prolylglutamic acid Proteins 0.000 description 1
- 238000003908 quality control method Methods 0.000 description 1
- 108700012613 rat Tafazzin Proteins 0.000 description 1
- 238000009790 rate-determining step (RDS) Methods 0.000 description 1
- 239000011541 reaction mixture Substances 0.000 description 1
- 108700005467 recombinant KCB-1 Proteins 0.000 description 1
- 230000001105 regulatory effect Effects 0.000 description 1
- 230000004044 response Effects 0.000 description 1
- 102000000568 rho-Associated Kinases Human genes 0.000 description 1
- 108010041788 rho-Associated Kinases Proteins 0.000 description 1
- 229920002477 rna polymer Polymers 0.000 description 1
- 238000009738 saturating Methods 0.000 description 1
- 238000006748 scratching Methods 0.000 description 1
- 230000002393 scratching effect Effects 0.000 description 1
- 230000011218 segmentation Effects 0.000 description 1
- 238000010206 sensitivity analysis Methods 0.000 description 1
- 238000002864 sequence alignment Methods 0.000 description 1
- 108010069117 seryl-lysyl-aspartic acid Proteins 0.000 description 1
- 229910052709 silver Inorganic materials 0.000 description 1
- 239000004332 silver Substances 0.000 description 1
- 239000011780 sodium chloride Substances 0.000 description 1
- 238000007619 statistical method Methods 0.000 description 1
- 229940115920 streptococcus dysgalactiae Drugs 0.000 description 1
- 230000009469 supplementation Effects 0.000 description 1
- 230000001502 supplementing effect Effects 0.000 description 1
- 230000004083 survival effect Effects 0.000 description 1
- 238000003786 synthesis reaction Methods 0.000 description 1
- 108010031491 threonyl-lysyl-glutamic acid Proteins 0.000 description 1
- 239000003104 tissue culture media Substances 0.000 description 1
- 231100000331 toxic Toxicity 0.000 description 1
- 230000002588 toxic effect Effects 0.000 description 1
- 231100000419 toxicity Toxicity 0.000 description 1
- 230000001988 toxicity Effects 0.000 description 1
- 238000013518 transcription Methods 0.000 description 1
- 230000035897 transcription Effects 0.000 description 1
- 108091006107 transcriptional repressors Proteins 0.000 description 1
- 239000012096 transfection reagent Substances 0.000 description 1
- 230000009261 transgenic effect Effects 0.000 description 1
- 230000001052 transient effect Effects 0.000 description 1
- 238000009966 trimming Methods 0.000 description 1
- LENZDBCJOHFCAS-UHFFFAOYSA-N tris Chemical compound OCC(N)(CO)CO LENZDBCJOHFCAS-UHFFFAOYSA-N 0.000 description 1
- 108010038745 tryptophylglycine Proteins 0.000 description 1
- 108010012567 tyrosyl-glycyl-glycyl-phenylalanyl Proteins 0.000 description 1
- 108010051110 tyrosyl-lysine Proteins 0.000 description 1
- 241001496653 uncultured Termite group 1 bacterium phylotype Rs-D17 Species 0.000 description 1
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/85—Vectors or expression systems specially adapted for eukaryotic hosts for animal cells
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/195—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from bacteria
- C07K14/315—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from bacteria from Streptococcus (G), e.g. Enterococci
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/113—Non-coding nucleic acids modulating the expression of genes, e.g. antisense oligonucleotides; Antisense DNA or RNA; Triplex- forming oligonucleotides; Catalytic nucleic acids, e.g. ribozymes; Nucleic acids used in co-suppression or gene silencing
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/85—Vectors or expression systems specially adapted for eukaryotic hosts for animal cells
- C12N15/86—Viral vectors
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/87—Introduction of foreign genetic material using processes not otherwise provided for, e.g. co-transformation
- C12N15/90—Stable introduction of foreign DNA into chromosome
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/87—Introduction of foreign genetic material using processes not otherwise provided for, e.g. co-transformation
- C12N15/90—Stable introduction of foreign DNA into chromosome
- C12N15/902—Stable introduction of foreign DNA into chromosome using homologous recombination
- C12N15/907—Stable introduction of foreign DNA into chromosome using homologous recombination in mammalian cells
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N5/00—Undifferentiated human, animal or plant cells, e.g. cell lines; Tissues; Cultivation or maintenance thereof; Culture media therefor
- C12N5/06—Animal cells or tissues; Human cells or tissues
- C12N5/0602—Vertebrate cells
- C12N5/0696—Artificially induced pluripotent stem cells, e.g. iPS
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N5/00—Undifferentiated human, animal or plant cells, e.g. cell lines; Tissues; Cultivation or maintenance thereof; Culture media therefor
- C12N5/10—Cells modified by introduction of foreign genetic material
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/14—Hydrolases (3)
- C12N9/16—Hydrolases (3) acting on ester bonds (3.1)
- C12N9/22—Ribonucleases RNAses, DNAses
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07H—SUGARS; DERIVATIVES THEREOF; NUCLEOSIDES; NUCLEOTIDES; NUCLEIC ACIDS
- C07H21/00—Compounds containing two or more mononucleotide units having separate phosphate or polyphosphate groups linked by saccharide radicals of nucleoside groups, e.g. nucleic acids
- C07H21/02—Compounds containing two or more mononucleotide units having separate phosphate or polyphosphate groups linked by saccharide radicals of nucleoside groups, e.g. nucleic acids with ribosyl as saccharide radical
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07H—SUGARS; DERIVATIVES THEREOF; NUCLEOSIDES; NUCLEOTIDES; NUCLEIC ACIDS
- C07H21/00—Compounds containing two or more mononucleotide units having separate phosphate or polyphosphate groups linked by saccharide radicals of nucleoside groups, e.g. nucleic acids
- C07H21/04—Compounds containing two or more mononucleotide units having separate phosphate or polyphosphate groups linked by saccharide radicals of nucleoside groups, e.g. nucleic acids with deoxyribosyl as saccharide radical
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/10—Type of nucleic acid
- C12N2310/20—Type of nucleic acid involving clustered regularly interspaced short palindromic repeats [CRISPRs]
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2510/00—Genetically modified cells
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2740/00—Reverse transcribing RNA viruses
- C12N2740/00011—Details
- C12N2740/10011—Retroviridae
- C12N2740/15011—Lentivirus, not HIV, e.g. FIV, SIV
- C12N2740/15041—Use of virus, viral particle or viral elements as a vector
- C12N2740/15043—Use of virus, viral particle or viral elements as a vector viral genome or elements thereof as genetic vector
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2800/00—Nucleic acids vectors
- C12N2800/90—Vectors containing a transposable element
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2830/00—Vector systems having a special element relevant for transcription
- C12N2830/001—Vector systems having a special element relevant for transcription controllable enhancer/promoter combination
- C12N2830/002—Vector systems having a special element relevant for transcription controllable enhancer/promoter combination inducible enhancer/promoter combination, e.g. hypoxia, iron, transcription factor
- C12N2830/003—Vector systems having a special element relevant for transcription controllable enhancer/promoter combination inducible enhancer/promoter combination, e.g. hypoxia, iron, transcription factor tet inducible
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Genetics & Genomics (AREA)
- Engineering & Computer Science (AREA)
- Chemical & Material Sciences (AREA)
- Organic Chemistry (AREA)
- Biomedical Technology (AREA)
- Biotechnology (AREA)
- Wood Science & Technology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Zoology (AREA)
- General Engineering & Computer Science (AREA)
- Molecular Biology (AREA)
- Biochemistry (AREA)
- General Health & Medical Sciences (AREA)
- Microbiology (AREA)
- Biophysics (AREA)
- Plant Pathology (AREA)
- Physics & Mathematics (AREA)
- Cell Biology (AREA)
- Mycology (AREA)
- Transplantation (AREA)
- Developmental Biology & Embryology (AREA)
- Virology (AREA)
- Medicinal Chemistry (AREA)
- Gastroenterology & Hepatology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Enzymes And Modification Thereof (AREA)
- Saccharide Compounds (AREA)
- Preparation Of Compounds By Using Micro-Organisms (AREA)
- Peptides Or Proteins (AREA)
Abstract
반복부 서열이 결핍된 TALEN 또는 Cas9를 이용하여 세포에서 게놈을 조작하는 방법을 제공한다.
Description
관련 출원에 관한 데이터
본 출원은 2013년 7월 26일 출원된 미국 가특허 출원 번호 61/858,866을 우선권 주장하며, 이 출원은 그 전문이 모든 목적을 위해 본원에서 참조로 포함된다.
정부 이익에 관한 언급
본 발명은 국립 인간 게놈 연구소 게놈 과학 우수 센터(National Human Genome Research Center for Excellence in Genomics Science)로부터의 P50 HG003170 하의 정부 지원으로 만들어졌다. 정부가 본 발명에 대한 특정 권리를 가진다.
서열-특이 뉴클레아제를 통한 게놈 편집은 공지되어 있다. 그의 전문이 본원에 참조로 포함된 참고 문헌 1, 2, 및 3을 참조할 수 있다. 게놈 중 뉴클레아제-매개 이중 가닥 DNA (dsDNA) 절단은, 빈번하게는 비-특이 삽입 및 결실 (indels)의 도입을 결과로서 초래하는 비-상동성 단부 연결 (NHEJ), 또는 수복 주형으로서 상동성 가닥을 도입하는 상동성 지정 수복 (HDR)이라는 2가지 주요 기전에 의해 수복될 수 있다. 그 전문이 본원에 참조로 포함된 참고 문헌 4를 참조할 수 있다. 서열-특이 뉴클레아제가 원하는 돌연변이를 함유하는 상동성 도너 DNA를 따라 전달될 때, 유전자 표적화 효율은 단지 도너 구축물 단독인 것과 비교하였을 때 1,000배 증가되어 있다. 그 전문이 본원에 참조로 포함된 참고 문헌 5를 참조할 수 있다. DNA 도너로서 단일 가닥 올리고데옥시리보뉴클레오티드 ("ssODN")를 사용하는 것이 보고된 바 있다. 그 전문이 본원에 참조로 포함된 참고 문헌 21 및 22를 참조할 수 있다.
유전자 편집 도구가 크게 진보했음에도 불구하고, 인간 유도 다능성 줄기 세포 ("hiPSC") 조작에서 맞춤식 조작된 뉴클레아제 사용과 관련해서는 많은 도전 과제와 의문이 여전히 남아있다. 첫째, 그의 디자인이 단순함에도 불구하고, 전사 활성인자-유사 효과기 뉴클레아제 (TALEN)는 반복 가변 이잔기 (RVD) 도메인의 일렬 카피를 포함하는 특정 DNA 서열을 표적화한다. 그 전문이 본원에 참조로 포함된 참고 문헌 6을 참조할 수 있다. RVD의 모듈식 성질을 통해 TALEN 디자인이 간소화되었지만, 그의 반복 서열은 그의 DNA 구축물을 합성하는 방법을 복잡하게 만들고 (그 전문이 본원에 참조로 포함된 참고 문헌 2, 9, 및 15-19를 참조할 수 있다), 이는 또한 렌티바이러스 유전자 전달 비히클을 이용하는 그의 사용을 손상시킨다. 그 전문이 본원에 참조로 포함된 참고 문헌 13을 참조할 수 있다.
현 관행상, NHEJ 및 HDR은 빈번하게는 별개의 검정법을 시용하여 평가된다. NHEJ를 검정하는 데에는 대개 미스매치-고감도 엔도뉴클레아제 검정법 (그 전문이 본원에 참조로 포함된 참고 문헌 14를 참조할 수 있다)이 사용되지만, 상기 방법의 정량적 정확성은 가변적이고, 감도는 ~3% 초과로 NHEJ 빈도에 한정된다. 그 전문이 본원에 참조로 포함된 참고 문헌 15를 참조할 수 있다. HDR은 빈번하게는, 완전히 다르고, 대개는 번잡한 방법인 클로닝 및 시퀀싱에 의해 평가된다. 일부 세포 유형, 예컨대, U2OS 및 K562의 경우에는 대략 50%로 편집 빈도가 높은 것으로 빈번하게 보고되어 있지만 (그 전문이 본원에 참조로 포함된 참고 문헌 12 및 14를 참조할 수 있다), hiPSC에서 빈도는 일반적으로 더 낮기 때문에, 감도가 여전히 문제가 된다. 그 전문이 본원에 참조로 포함된 참고 문헌 10을 참조할 수 있다. 최근, hiPSC 및 hESC에서 TALEN을 사용하는 경우, 편집 빈도가 높은 것으로 보고되었고 (그 전문이 본원에 참조로 포함된 참고 문헌 9를 참조할 수 있다), CRISPR Cas9-gRNA 시스템을 사용하는 경우에는 빈도가 훨씬 더 높은 것으로 보고되었다 (그 전문이 본원에 참조로 포함된 참고 문헌 16-19를 참조할 수 있다). 그러나, 다른 부위에서의 편집률은 매우 다른 것으로 보이며 (그 전문이 본원에 참조로 포함된 참고 문헌 17을 참조할 수 있다), 편집은 종종 일부 부위에서는 전혀 검출되지 않는다 (그 전문이 본원에 참조로 포함된 참고 문헌 20을 참조할 수 있다).
박테리아 및 고세균 CRISPR-Cas 시스템은 침입 외래 핵산 내에 존재하는 상보적인 서열의 분해를 지시하는, Cas 단백질과 함께 복합된 짧은 가이드 RNA에 의존한다. 문헌 [Deltcheva, E. et al. CRISPR RNA maturation by trans-encoded small RNA 및 host factor RNase III. Nature 471, 602-607 (2011)]; [Gasiunas, G., Barrangou, R., Horvath, P. & Siksnys, V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proceedings of the National Academy of Sciences of the United States of America 109, E2579-2586 (2012)]; [Jinek, M. et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science 337, 816-821 (2012)]; [Sapranauskas, R. et al. The Streptococcus thermophilus CRISPR/Cas systems provides immunity in Escherichia coli. Nucleic acids research 39, 9275-9282 (2011)]; 및 [Bhaya, D., Davison, M. & Barrangou, R. CRISPR-Cas systems in bacteria and archaea: versatile small RNAs for adaptive defense and regulation. Annual review of genetics 45, 273-297 (2011)]을 참조할 수 있다. 최근 S. 피오게네스(S. pyogenes) II형 CRISPR의 시험관내 재구성을 통해 보통 트랜스 코딩된 tracrRNA ("트랜스-활성 CRISPR RNA")에 융합된 crRNA ("CRISPR RNA")가 Cas9 단백질을 지시하여 상기 crRNA에 매칭되는 표적 DNA를 서열 특이적으로 절단하는 데에는 충분하다는 것이 입증되었다. 표적 부위에 상동성인 gRNA가 발현되면, Cas9가 동원되고, 표적 DNA가 분해된다. 문헌 [H. Deveau et al., Phage response to CRISPR-encoded resistance in Streptococcus thermophilus. Journal of Bacteriology 190, 1390 (Feb, 2008)]을 참조할 수 있다.
본 개시내용의 측면은 세포, 예컨대, 체세포 또는 줄기 세포를 유전적으로 변형시키기 위한, 변형된 전사 활성인자-유사 효과기 뉴클레아제 (TALEN)의 용도에 관한 것이다. TALEN은 반복부 서열을 포함하는 것으로 알려져 있다. 본 개시내용의 측면은 반복부 서열 100 bp 이상이 결핍된 TALEN을 세포 내로 도입하는 단계를 포함하고, 여기서, 세포에서 TALEN은 표적 DNA를 절단하고, 세포는 비상동성 단부 연결을 거치고, 이로써 변경된 DNA가 생산되는 것인, 세포에서 표적 DNA를 변경시키는 방법에 관한 것이다. 특정 측면에 따라, 원하는 길이의 반복부 서열이 TALEN으로부터 제거될 수 있다. 특정 측면에 따라, TALEN에는 특정의 원하는 길이의 반복부 서열이 없다. 특정 측면에 따라, 원하는 길이의 반복부 서열이 제거된 TALEN을 제공한다. 특정 측면에 따라, TALEN을 변형시켜 원하는 길이의 반복부 서열을 제거한다. 특정 측면에 따라, TALEN을 조작하여 원하는 길이의 반복부 서열을 제거한다.
본 개시내용의 측면은 세포에서 반복부 서열 100 bp 이상이 결핍된 TALEN 및 도너 핵산 서열을 조합하는 단계를 포함하고, 여기서, 세포에서 TALEN은 표적 DNA를 절단하고, 도너 핵산 서열은 DNA 내로 삽입되는 것인, 세포에서 표적 DNA를 변경시키는 방법에 관한 것이다. 본 개시내용의 측면은 반복부 서열 100 bp 이상이 결핍된 TALEN을 코딩하는 핵산 서열을 포함하는 바이러스에 관한 것이다. 본 개시내용의 측면은 반복부 서열 100 bp 이상이 결핍된 TALEN을 코딩하는 핵산 서열을 포함하는 세포에 관한 것이다. 본원에 기술된 특정 측면에 따라, TALEN에는 반복부 서열 100 bp 이상, 90 bp 이상, 80 bp 이상, 70 bp 이상, 60 bp 이상, 50 bp 이상, 40 bp 이상, 30 bp 이상, 20 bp 이상, 19 bp 이상, 18 bp 이상, 17 bp 이상, 16 bp 이상, 15 bp 이상, 14 bp 이상, 13 bp 이상, 12 bp 이상, 11 bp 이상, 또는 10 bp 이상이 결핍되어 있다.
본 개시내용의 측면은 엔도뉴클레아제, DNA 폴리머라제, DNA 리가제, 엑소뉴클레아제, 반복 가변 이잔기 도메인을 코딩하는 복수 개의 핵산 이량체 블록 및 엔도뉴클레아제 절단 부위를 포함하는 TALE-N/TF 골격 벡터를 조합하는 단계, 엔도뉴클레아제를 활성화시켜 엔도뉴클레아제 절단 부위에서 TALE-N/TF 골격 벡터를 절단하여 제1 단부 및 제2 단부를 제조하는 단계, 엑소뉴클레아제를 활성화시켜 TALE-N/TF 골격 벡터 및 복수 개의 핵산 이량체 블록 상에 3' 및 5' 오버행을 형성하고, TALE-N/TF 골격 벡터 및 복수 개의 핵산 이량체 블록을 원하는 순서로 어닐링하는 단계, 및 DNA 폴리머라제 및 DNA 리가제를 활성화시켜 TALE-N/TF 골격 벡터 및 복수 개의 핵산 이량체 블록을 연결시키는 단계를 포함하는, TALE 제조에 관한 것이다. 통상의 기술자는 본 개시내용에 기초하여 적합한 엔도뉴클레아제, DNA 폴리머라제, DNA 리가제, 엑소뉴클레아제, 반복 가변 이잔기 도메인을 코딩하는 핵산 이량체 블록 및 TALE-N/TF 골격 벡터를 쉽게 확인할 수 있을 것이다.
본 개시내용의 측면은 (a) 줄기 세포 내로 표적 DNA에 상보적이고, 효소를 표적 DNA로 가이드하는 RNA를 코딩하는 제1 외래 핵산을 도입하는 단계로서, 여기서, RNA 및 효소가 표적 DNA에 대한 공동 국재화 복합체의 구성원인 것인 단계, 줄기 세포 내로 도너 핵산 서열을 코딩하는 제2 외래 핵산을 도입하여 줄기 세포에서 변경된 DNA를 제조하는 단계로서, 여기서, RNA 및 도너 핵산 서열이 발현되고, 여기서, RNA 및 효소는 표적 DNA로 공동 국재화되고, 효소는 표적 DNA를 절단하고, 도너 핵산은 표적 DNA 내로 삽입되는 것인 단계를 포함하는, 표적 DNA에 상보적인 RNA와 공동 국재화 복합체를 형성하고, 부위 특이적인 방식으로 표적 DNA를 절단하는 효소를 발현하는 줄기 세포에서 표적 DNA를 변경시키는 방법에 관한 것이다.
본 개시내용의 측면은 표적 DNA에 상보적인 RNA와 공동 국재화 복합체를 형성하고, 부위 특이적인 방식으로 표적 DNA를 절단하는 효소를 코딩하는 제1 외래 핵산을 포함하는 줄기 세포에 관한 것이다.
본 개시내용의 측면은 표적 DNA에 상보적인 RNA와 공동 국재화 복합체를 형성하고, 부위 특이적인 방식으로 표적 DNA를 절단하는 효소를 코딩하는 제1 외래 핵산을 포함하고, 효소의 발현을 촉진시키는 유도성 프로모터를 포함하는 것인 세포에 관한 것이다. 이러한 방식으로, 발현은 조절될 수 있고, 예를 들어, 개시될 수 있고, 종결될 수 있다.
본 개시내용의 측면은 표적 DNA에 상보적인 RNA와 공동 국재화 복합체를 형성하고, 부위 특이적인 방식으로 표적 DNA를 절단하는 효소를 코딩하는 제1 외래 핵산을 포함하는 세포로서, 여기서, 제1 외래 핵산은 제거 효소, 예컨대, 트랜스포사제를 사용하여 세포의 게놈 DNA로부터 제거가능한 것인, 세포에 관한 것이다.
본 개시내용의 측면은 (a) 세포 내로 도너 핵산 서열을 코딩하는 제1 외래 핵산을 도입하는 단계, 세포 주변 배지로부터 세포 내로 표적 DNA에 상보적이고, 효소를 표적 DNA로 가이드하는 RNA를 도입하여 세포에서 변경된 DNA를 제조하는 단계로서, 여기서, RNA 및 효소가 표적 DNA에 대한 공동 국재화 복합체의 구성원이고, 여기서, 도너 핵산 서열이 발현되고, 여기서, RNA 및 효소는 표적 DNA로 공동 국재화되고, 효소는 표적 DNA를 절단하고, 도너 핵산은 표적 DNA 내로 삽입되는 것인 단계를 포함하는, 표적 DNA에 상보적인 RNA와 공동 국재화 복합체를 형성하고, 부위 특이적인 방식으로 표적 DNA를 절단하는 효소를 발현하는 세포에서 표적 DNA를 변경시키는 방법에 관한 것이다.
본 개시내용의 측면은 줄기 세포를 유전적으로 변형시키기 위한 RNA에 의해 가이드되는 DNA 결합 단백질의 용도에 관한 것이다. 한 측면에서, 줄기 세포는 RNA에 의해 가이드되는 DNA 결합 단백질을 코딩하는 핵산을 포함하도록 유전적으로 변형되고, 줄기 세포는 RNA에 의해 가이드되는 DNA 결합 단백질을 발현한다. 특정 측면에 따라, 특이 돌연변이를 도입하기 위한 도너 핵산은 변형된 TALEN 또는 RNA에 의해 가이드되는 DNA 결합 단백질을 사용하여 게놈 편집을 위해 최적화된다.
본 개시내용의 측면은 하나 이상의 가이드 RNA (리보핵산)를 사용하여 줄기 세포에 의해 발현된 뉴클레아제 활성을 가지는 효소, 예컨대, 뉴클레아제 활성을 가지는 DNA 결합 단백질을 DNA (데옥시리보핵산) 상의 표적 위치로 유도하는 것으로서, 여기서, 효소는 DNA를 절단하고, 외인성 도너 핵산이 예컨대, 상동성 재조합에 의해 DNA 내로 삽입되는 것인, 줄기 세포에서의 DNA 변형, 예컨대, DNA의 다중 변형에 관한 것이다. 본 개시내용의 측면은 줄기 세포 상에서 DNA 변형 단계를 사이클링 또는 반복하여 세포 내에서 DNA의 다중 변형을 가지는 줄기 세포를 제조하는 것을 포함한다. 변형은 외인성 도너 핵산의 삽입을 포함할 수 있다.
다중 외인성 핵산 삽입은 복수 개의 RNA를 코딩하는 핵산 및 복수 개의 외인성 도너 핵산을 예컨대, 공동 형질전환에 의해 효소를 발현하는 줄기 세포 내로 도입하는 단일 단계로서, 여기서, RNA는 발현되고, 여기서, 복수 개의 각 RNA는 효소를 DNA의 특정 부위로 가이드하고, 효소는 DNA를 절단하고, 복수 개의 외인성 핵산 중 하나는 절단 부위에서 DNA 내로 삽입되는 것인 단계에 의해 달성될 수 있다. 따라서, 본 측면에 따라, 세포에서의 DNA의 다수의 변경 또는 변형은 단일 사이클로 형성된다.
다중 외인성 핵산 삽입은 하나 이상의 RNA 또는 복수 개의 RNA를 코딩하는 하나 이상의 핵산 및 하나 이상의 외인성 핵산 또는 복수 개의 외인성 핵산을 효소를 발현하는 줄기 세포 내로 도입하는 반복 단계 또는 사이클로서, 여기서, RNA는 발현되고, 효소를 DNA의 특정 부위로 가이드하고, 효소는 DNA를 절단하고, 외인성 핵산은 절단 부위에서 DNA 내로 삽입되어 줄기 세포 내에서 DNA 내로의 다수의 변경 또는 외인성 DNA의 삽입을 가지는 세포를 생성하는 것인 단계 또는 사이클에 의해 세포에서 달성될 수 있다. 따라서, 한 측면에 따라, 효소를 발현하는 줄기 세포는 예컨대, 세포 내로 효소를 코딩하고, 줄기 세포에 의해 발현될 수 있는 핵산을 도입함으로써 효소를 발현하도록 유전적으로 변경되었다. 이러한 방식으로, 본 개시내용의 측면은 효소를 발현하는 줄기 세포 내로 RNA를 도입하는 단계, 줄기 세포 내로 외인성 도너 핵산을 도입하는 단계, RNA를 발현하는 단계, RNA, 효소, 및 DNA의 공동 국재화 복합체를 형성하는 단계, 효소에 의해 DNA를 효소적으로 절단하는 단계, 및 도너 핵산을 DNA 내로 삽입하는 단계를 사이클링하는 것을 포함한다. 상기 단계를 사이클링 또는 반복함으로써 줄기 세포를 다중 유전자좌에서 다중으로 유전적으로 변형시킬 수 있고, 즉, 다중의 유전적 변형을 가지는 줄기 세포를 생성할 수 있다.
특정 측면에 따라, 본 개시내용의 범주 내의 DNA 결합 단백질 또는 효소는 가이드 RNA와 복합체를 형성하는 단백질을 포함하고, 가이드 RNA는 상기 복합체를 이중 가닥 DNA 서열로 가이드하며, 여기서, 복합체는 DNA 서열에 결합한다. 한 측면에 따라, 효소는 RNA에 의해 가이드되는 DNA 결합 단백질, 예컨대, DNA에 결합하고, RNA에 의해 가이드되는, II형 CRISPR 시스템의 RNA에 의해 가이드되는 DNA 결합 단백질일 수 있다. 한 측면에 따라, RNA에 의해 가이드되는 DNA 결합 단백질은 Cas9 단백질이다.
본 개시내용의 상기 측면은 이중 가닥 DNA에의, 또는 그와의 RNA 및 DNA 결합 단백질의 공동 국재화로 지칭될 수 있다. 이러한 방식으로, DNA 결합 단백질-가이드 RNA 복합체는 다중으로 유전적 변형된 줄기 세포, 예컨대, 외인성 도너 DNA가 다중으로 삽입된 줄기 세포를 생성하기 위해 이중 가닥 DNA의 다중 부위를 절단하는 데 사용될 수 있다.
특정 측면에 따라, (a) 줄기 세포 내로 표적 DNA에 상보적이고, 효소를 표적 DNA로 가이드하는 하나 이상의 RNA를 코딩하는 제1 외래 핵산을 도입하는 단계로서, 여기서, 하나 이상의 RNA 및 효소가 표적 DNA에 대한 공동 국재화 복합체의 구성원인 것인 단계, 줄기 세포 내로 하나 이상의 도너 핵산 서열을 코딩하는 제2 외래 핵산을 도입하여 줄기 세포에서 변경된 DNA를 제조하는 단계로서, 여기서, 하나 이상의 RNA 및 하나 이상의 도너 핵산 서열이 발현되고, 여기서, 하나 이상의 RNA 및 효소는 표적 DNA로 공동 국재화되고, 효소는 표적 DNA를 절단하고, 도너 핵산은 표적 DNA 내로 삽입되는 것인 단계, 및 단계 (a)를 다회에 걸쳐 반복하여 줄기 세포에서 DNA를 다중으로 변경시키는 단계를 포함하는, 표적 DNA에 상보적인 RNA와 공동 국재화 복합체를 형성하고, 부위 특이적인 방식으로 표적 DNA를 절단하는 효소를 발현하는 줄기 세포에서 표적 DNA를 다중으로 변경시키는 방법을 제공한다.
한 측면에 따라, RNA는 약 10 내지 약 500개의 뉴클레오티드이다. 한 측면에 따라, RNA는 약 20 내지 약 100개의 뉴클레오티드이다.
한 측면에 따라, 하나 이상의 RNA는 가이드 RNA이다. 한 측면에 따라, 하나 이상의 RNA는 tracrRNA-crRNA 융합체이다.
한 측면에 따라, DNA는 게놈 DNA, 미토콘드리아 DNA, 바이러스 DNA, 또는 외인성 DNA이다.
한 측면에 따라, 세포는 효소를 사용하여 쉽게 제거될 수 있는 벡터를 이용하여 DNA 결합 효소를 코딩하는 핵산을 가역적으로 포함하도록 유전적으로 변형될 수 있다. 유용한 벡터 방법은 통상의 기술자에게 공지되어 있고, 렌티바이러스, 다에노 관련 바이러스, 뉴클레아제 및 인테그라제 매개 표적 삽입 방법 및 트랜스포존 매개 삽입 방법을 포함한다. 한 측면에 따라, 예컨대, 카세트 또는 벡터에 의해 부가된 DNA 결합 효소를 코딩하는 핵산은 예를 들어, 그 전체로 카세트 및 벡터와 함께, 및 게놈 DNA 중 상기 핵산, 카세트 또는 벡터의 일부를 남기지 않고 제거될 수 있다. 상기와 같은 제거는, 게놈이 핵산, 카세트 또는 벡터의 부가 전과 동일하기 때문에 관련 기술분야에서 "흔적 없는" 제거로 지칭된다. 삽입 및 흔적 없는 제거에 대한 한 예시적인 실시양태는 시스템 바이오사이언시즈(System Biosciences)로부터 상업적으로 이용가능한 피기백(PiggyBac) 벡터이다.
본 발명의 특정 실시양태의 추가의 특징 및 이점은 하기 실시양태에 대한 상세한 설명 및 그의 도면에서, 및 청구범위로부터 더욱 충분하게 자명해질 것이다.
본 실시양태의 상기 및 다른 특징 및 이점은 첨부된 도면과 함께 하기의 예시적인 실시양태에 관한 상세한 설명으로부터 더욱 충분히 이해될 것이다:
도 1은 인간 체세포 및 줄기 세포에서의 리TALEN의 기능적 시험에 관한 것이다.
(a) 게놈 표적화 효율을 시험하기 위한 실험 디자인의 도식적 대표도. 종결 코돈 및 AAVS1 유전자좌로부터 유래된 68 bp 게놈 단편의 삽입에 의해 게놈적으로 통합된 GFP 코딩 서열을 파괴한다 (하단). tGFP 도너와의 뉴클레아제-매개 상동성 재조합에 의한 GFP 서열의 복원 (상단)을 통해 FACS에 의해 정량화될 수 있는 GFP+ 세포를 수득한다. 리TALEN 및 TALEN은 AAVS1 단편 내의 동일한 서열을 표적화한다.
(b) FACS에 의해 측정된, tGFP 도너만이 단독으로, tGFP 도너와 함께 TALEN, 및 tGFP 도너와 함께 리TALEN이 표적 유전자좌에 도입된 GFP+ 세포의 비율(%)을 도시한 막대 그래프. (N=3, 오차 막대=SD). 대표 FACS 플롯이 하기에 제시되어 있다.
(c) 천연 AAVS1 유전자좌에 대한 표적화 전략을 도시한 도식적 개요. 스플라이싱 억셉터 (SA)- 2A (자가-절단 펩티드), 퓨로마이신 저항성 유전자 (PURO) 및 GFP를 함유하는 도너 플라스미드가 기술되었다 (그 전문이 본원에 참조로 포함된 참고 문헌 10을 참조할 수 있다). 성공적인 편집 이벤트를 검출하는 데 사용된 PCR 프라이머의 위치는 청색 화살표로 표시되어 있다.
(d) 2주 동안 퓨로마이신 (0.5ug/mL)을 이용하여 PGP1 hiPSC의 성공적으로 표적화된 클론을 선별하였다. 대표적인 GFP+ 클론 3개에 대한 현미경 영상이 제시되어 있다. 세포를 또한 다능성 마커 TRA-1-60에 대하여 염색하였다. 스케일 바: 200 ㎛.
(e) 상기의 단일클론 GFP+ hiPSC 클론에서 수행된 PCR 검정법을 통해 도너 카세트가 AAVS1 부위에 성공적으로 삽입된 반면 (레인 1,2,3), 단순 hiPSC는 성공적인 삽입에 관한 어떤 증거도 보이지 않았다는 것 (레인 C)이 입증되었다.
도 2는 iPSC 중 CCR5 상에서의 리TALEN 및 Cas9-gRNA 게놈 표적화 효율 비교에 관한 것이다.
(a) 게놈 조작 실험 디자인의 도식적 대표도. 리TALEN 쌍 또는 Cas9-gRNA 표적화 부위에서 게놈 DNA에 대한 2 bp 미스매치를 보유하는 90mer ssODN을 리TALEN 또는 Cas9-gRNA 구축물과 함께 PGP1 hiPSC 내로 전달하였다. 도면에서 뉴클레아제의 절단 부위는 적색 화살표로 표시되어 있다.
(b) 리TALEN 쌍 (CCR5 #3) 및 ssODN, 또는 Cas9-gRNA 및 ssODN에 대한 HDR 및 NHEJ 효율에 관한 심층 서열분석 분석. hiPSC의 게놈 중의 변경을 GEAS에 의한 고처리량 서열 데이터로부터 분석하였다. 상단: HDR은 ssODN의 중앙부 내로 구축된 2 bp 점 돌연변이를 함유하는 리드의 분율로부터 정량화하고 (청색), NHEJ 활성은 게놈 내의 각 특이 위치에서의 결실 (회색)/삽입 (적색)의 분율로부터 정량화하였다. 리TALEN 및 ssODN 그래프의 경우, 녹색 점선을 플롯팅하여 두 리TALEN 결합 부위의 중앙부에 대해 상대적인 위치로 -26 bp 내지 +26 bp 위치인 리TALEN 쌍의 결합 부위의 외곽 경계를 마킹하였다. Cas9-gRNA 및 ssODN 그래프의 경우, 녹색 점선은 PAM 서열에 대해 상대적인 위치로 -20 내지 -1 bp 위치인 gRNA 표적화 부위의 외곽 경계를 마킹한다. 하단: 상기 명시된 바와 같이 처리된 전체 NHEJ 집단으로부터 분석된 hiPSC 중의 결실/삽입 크기 분포.
(c) PGP1 hiPSC에서의 리TALEN 및 Cas9-gRNA 표적화 CCR5의 게놈 편집 효율.
상단: CCR5에서 표적화된 게놈 편집 부위에 관한 도식적 대표도. 15개의 표적화 부위가 하기에 청색 화살표로 도시되어 있다. 각 부위에 대하여 세포를 리TALEN, 및 게놈 DNA에 대한 2 bp 미스매치를 보유하는 그의 상응하는 ssODN 도너로 이루어진 쌍으로 공동 형질감염시켰다. 형질감염 후 6일 경과하였을 때 게놈 편집 효율을 검정하였다. 유사하게, 15개의 Cas9-gRNA를 그의 상응하는 ssODN을 이용하여 개별적으로 PGP1-hiPSC 내로 형질감염시켜 같은 15개의 부위를 표적화하고, 형질감염 후 6일 경과하였을 때 효율을 분석하였다. 하단: PGP1 hiPSC에서의 리TALEN 및 Cas9-gRNA 표적화 CCR5의 게놈 편집 효율. 패널 1 및 2는 리TALEN에 의해 매개된 NHEJ 및 HDR 효율을 나타낸다. 패널 3 및 4는 Cas9-gRNA에 의해 매개된 NHEJ 및 HDR 효율을 나타낸다. NHEJ 비율은 표적화 부위에 결실 또는 삽입을 보유하는 게놈 대립 유전자의 빈도에 의해 계산하였고; HDR 비율은 2 bp 미스매치를 보유하는 게놈 대립 유전자의 빈도에 의해 계산하였다. 패널 5는 엔코드(ENCODE) 데이터베이스로부터의 hiPSC 세포주의 DNaseI HS 프로파일이다 (듀크(Duke) DNase HS, iPS NIHi7 DS). 특히, 다른 패널의 스케일은 상이하다.
(f) 3개의 표적화된 hiPSC 콜로니로부터의 PCR 앰플리콘의 생어(Sanger) 서열분석을 통해 게놈-삽입 경계에 예상 DNA 염기가 존재한다는 것을 확인하였다. M: DNA 래더. C: 단순 hiPSC 게놈 DNA를 포함하는 대조군.
도 3은 PGP1 hiPSC에서의 리TALEN 또는 Cas9-gRNA를 이용하는 ssODN-매개 HDR을 지배하는 기능적 파라미터에 관한 연구에 관한 것이다.
(a) PGP1 hiPSC를 리TALEN 쌍 (#3) 및 상이한 길이 (50, 70, 90, 110, 130, 150, 170 nts)의 ssODN으로 공동 형질감염시켰다. 모든 ssODN은 그의 서열 중간에 게놈 DNA에 대하여 동일한 2 bp 미스매치를 포함하였다. 90mer ssODN은 표적화된 게놈에서 최적의 HDR을 달성하였다. HDR, NHEJ 발생된 결실 및 삽입 효율 평가는 본원에 기술되어 있다.
(b) 각각이 중앙에 2 bp 미스매치 (A) 및 A로부터의 상이한 위치 상쇄부 (여기서, 상쇄부는 -30 bp→30 bp로 달랐다)에 추가의 2 bp 미스매치 (B)를 함유하는 리TALEN 쌍 #3에 상응하는 90mer ssODN을 사용하여 ssODN에 따른 상동성으로부터의 이탈의 효과를 시험하였다. 각 ssODN의 게놈 편집 효율은 PGP1 hiPSC에서 평가하였다. 하단의 막대 그래프는 표적화된 게놈에서의 오직 A만, 오직 B만, 및 A + B의 도입 빈도를 보여주는 것이다. HDR 비율은 중앙으로부터의 상동성 이탈 거리가 증가할수록 감소한다.
(c) 리TALEN 쌍 #3의 표적 부위로부터 다양한 거리 (-620 bp ~ 480 bp)를 두고 떨어져 있는 부위로 표적화되는 ssODN을 시험함으로써 돌연변이를 도입하는 데 ssODN이 배치될 수 있는 최대 거리를 평가하였다. 모든 ssODN은 그의 서열 한가운데에 2 bp 미스매치를 보유하였다. ssODN 미스매치가 리TALEN 쌍의 결합 부위의 한가운데로부터 40 bp만큼 떨어져 위치할 때 최소 HDR 효율 (<=0.06%)이 관찰되었다.
(d) PGP1 hiPSC를 Cas9-gRNA (AAVS1) 및 상이한 배향 (Oc: gRNA에 대해 상보적; On: gRNA에 대해 비상보적) 및 상이한 길이 (30, 50, 70, 90, 110 nt)의 ssODN으로 공동 형질감염시켰다. 모든 ssODN은 그의 서열 한가운데에 게놈 DNA에 대하여 동일한 2 bp 미스매치를 포함하였다. 70 mer Oc가 표적화된 게놈에서 최적의 HDR을 달성하였다.
도 4는 선별 없이 단일클론 게놈 편집된 hiPSC를 수득하기 위해 리TALEN 및 ssODN을 사용하는 것에 관한 것이다.
(a) 실험 타임라인.
(b) 도 2b에 기술된 NGS 플랫폼에 의해 평가된 리TALEN 쌍 및 ssODN (#3)의 게놈 조작 효율.
(c) 게놈 편집 이후의 단일클론 hiPSC 콜로니의 생어 서열분석 결과. 2 bp 이형접합성 유전자형 (CT/CT→TA/CT)이 PGP1-iPS-3-11, PGP1-iPS-3-13 콜로니의 게놈 내로 성공적으로 도입되었다.
(d) 표적화된 PGP1-iPS-3-11의 면역형광 염색. 세포를 다능성 마커 Tra-1-60 및 SSEA4에 대해 염색하였다.
(e) 단일클론 PGP1-iPS-3-11 세포로부터 생성된 테라토마 섹션의 헤마톡실린 및 에오신 염색.
도 5. 리TALE 디자인. (a) 리TALE-16.5에서의 단량체와 함께 원래의 TALE RVD 단량체로 이루어진 서열 정렬 (리TALE-M1→리TALE-M17). 원래의 서열로부터의 뉴클레오티드 변경은 회색으로 강조 표시되어 있다. (b) PCR에 의한 리TALE의 반복성 시험. 상단의 패널은 PCR 반응에서의 프라이머의 위치 및 리TALE/TALE의 구조를 도시한 것이다. 하단의 패널은 PCR 밴드를 도시한 것이고, 조건은 하기에 명시되어 있다. 원래의 TALE 주형 (우측 레인)과 함께 PCR 래더링이 제시되어 있다.
도 6. TALE 단일-인큐베이션 조립 (TASA) 조립 디자인 및 실시.
(a) TASA 조립을 위한 리TALE 이량체 블록의 라이브러리의 도식적 대표도. 2개의 RVD를 코딩하는 10개의 리TALE 이량체 블록의 라이브러리가 존재한다. 각 블록 내에서 16개의 이량체는 모두 RVD를 코딩하는 서열을 제외하면 동일한 DNA 서열을 공유한다; 다른 블록 중의 이량체는 독특한 서열을 가지지만, 그가 인접 블록과 32 bp의 중첩부를 공유하도록 디자인된다. 한 이량체 (블록6_AC)의 DNA 및 아미노산 서열은 우측에 열거되어 있다.
(b) TASA 조립의 도식적 대표도. 좌측 패널은 TASA 조립 방법을 도시한 것이다: 효소 혼합물/리TALE 블록/리TALE-N/TF 골격 벡터를 이용하여 원-포트 인큐베이션 반응을 수행한다. 반응 생성물은 박테리아 형질전환을 위해 직접 사용될 수 있다. 우측 패널은 TASA의 기전을 도시한 것이다. 37℃에서 엔도뉴클레아제에 의해 목표 벡터를 선형화하여 ccdB 역 선택 카세트를 절단하고; 블록의 단부 및 선형화된 벡터를 프로세싱하는 엑소뉴클레아제는 단편의 단부의 ssDNA 오버행을 노출시켜 블록 및 벡터 골격이 지정된 순서로 어닐링될 수 있도록 한다. 온도가 최대 50℃까지 상승하였을 때, 폴리머라제 및 리가제는 함께 작용하여 갭을 실링하고, 이로써, 형질전환을 위해 준비된 최종 구축물이 생성된다.
(c) 상이한 단량체 길이를 가지는 리TALE에 대한 TASA 조립 효율. 조립에 사용된 블록은 좌측에 도시되어 있고, 조립 효율은 우측에 제시되어 있다.
도 7. 렌티-리TALE의 기능성 및 서열 완전성.
도 8. GEAS의 감도 및 재현가능성.
(a) HDR 검출 한계의 정보 기반 분석. 리TALEN (#10)/ssODN인 데이터세트 전제하에, 예상되는 편집 (HDR)을 함유하는 리드를 확인하고, 이들 HDR 리드를 체계적으로 제거하여 편집 신호가 "희석된" 상이한 인공 데이터세트를 작성하였다. 제거율이 100, 99.8, 99.9, 98.9, 97.8, 89.2, 78.4, 64.9, 21.6, 10.8, 2.2, 1.1, 0.2, 0.1, 0.02, 및 0%인 HDR 리드를 포함하는 데이터세트는 HR 효율 범위가 0~0.67%인 인공 데이터세트로 작성되었다. 각각의 개별 데이터세트에 대하여, 배경 신호 (보라색), 및 표적화 부위에서 수득된 신호 (녹색)의 상호 정보 (MI)를 추정하였다. HDR 효율이 0.0014%를 초과할 때, 표적화 부위에서의 MI가 배경보다 현저히 더 높다. HDR 검출 한계는 0.0014% 내지 0.0071%인 것으로 추정되었다. MI 계산은 본원에 기술된 바와 같이 수행된다.
(b) 게놈 편집 평가 시스템의 재현가능성 시험. 플롯 쌍 (상단 및 하단)은 상기 명시된 리TALEN 쌍 및 세포 유형을 이용한 두 복제물의 HDR 및 NHEJ 평가 결과를 보여주는 것이다. 각 실험을 위해, 뉴클레오펙션, 표적화된 게놈 증폭, 심층 서열분석 및 데이터 분석을 독립적으로 수행하였다. 복제물의 게놈 편집 평가 편차는 √2(|HDR1-HDR2|)/((HDR+HDR2)/2) =ΔHDR/HDR 및 √2(|NHEJ1-NHEJ2|)/((NHEJ1+NHEJ2)/2) =ΔNHEJ/NHEJ로 계산하였고, 편차 결과는 하기 플롯에 열거되어 있다. 시스템의 평균 편차는 (19%+11%+4%+9%+10%+35%)/6=15%였다. 편차에 기여할 수 있는 인자로는 뉴클레오펙션하에 있는 세포의 상태, 뉴클레오펙션 효율, 및 서열분석 커버리지 및 품질을 포함한다.
도 9. CCR5 상에서의 리TALEN 및 Cas9-gRNA에 의한 NHEJ 및 HDR 효율의 통계하적 분석
(a) iPSC 중 동일 부위에서의 리TALEN에 의해 매개된 HR 및 NHEJ 효율의 상관관계 (r=0.91, P< 1X10-5).
(b) iPSC 중 동일 부위에서의 Cas9-gRNA에 의해 매개된 HR 및 NHEJ 효율의 상관관계 (r=0.74, P=0.002).
(c) iPSC에서의 Cas9-gRNA에 의해 매개된 NHEJ 효율 및 gRNA 표적화 부위의 Tm 온도의 상관관계 (r=0.52, P=0.04).
도 10. 게놈 편집 효율 및 후성적 상태의 상관관계 분석.
피어슨 상관관계를 사용하여 DNase I 감도와 게놈 조작 효율 (HR, NHEJ) 사이의 가능한 연관성을 연구하였다. 관찰된 상관관계를 임의 추출된 세트 (N=100000)와 비교하였다. 관찰된 상관관계가 모의 분포의 95번째 백분위수보다 더 높거나, 또는 5번째 백분위수보다 더 낮은 것은 잠재적으로 연관성이 있는 것으로 간주되었다. DNase I 감도와 NHEJ/HR 효율 사이에 어떤 뚜렷한 상관관계도 관찰되지 않았다.
도 11. ssODN-매개 게놈 편집에서의 상동성 쌍 형성의 영향.
(a) 도 3b에 기술된 실험에서, 중간 2 b 미스매치 (A)가 도입된 속도로 측정된 바, 전체 HDR은 제2 미스매치 B의 A로부터의 그의 거리가 증가함에 따라 감소하였다 (B에서 A까지의 상대적인 위치는 -30→30 bp로 다르다). B가 A로부터 단지 10 bp만큼 떨어져 있을 때 (-10 bp 내지 +10b)의 더 빠른 도입 속도는 dsDNA 절단에 인접한 게놈 DNA에 대한 ssODN의 쌍 형성이 덜 요구된다는 것을 반영하는 것일 수 있다.
(b) ssODN에 따른 유전자 전환 길이의 분포. A로부터의 B의 각 거리에서, HDR 이벤트 중 일부는 오직 A만을 도입하는 반면, 또 다른 일부는 A 및 B, 둘 모두를 도입한다. 상기 두 이벤트는 유전자 전환 트랙으로 해석될 수 있는데 (Elliott et al., 1998), 여기서, A+B 이벤트는 B 너머까지 확장된 긴 전환 트랙을 나타내고, 오직 A만의 이벤트는 B에 도달하지 못한 더 짧은 것을 나타낸다. 이러한 해석시, 올리고를 따른 양 방향의 유전자 전환 길이의 분포는 추정될 수 있다 (ssODN의 중간을 0으로, ssODN의 5' 단부쪽으로의 전환 트랙은 (-) 방향으로, 및 3' 단부 쪽으로의 것은 (+) 방향으로 정의된다). 유전자 전환 트랙은 그의 길이가 증가함에 따라 점진적으로 발생률이 감소하는데, 이 결과는 dsDNA 도너를 이용하였을 때 관찰된 유전자 전환 트랙 분포와 매우 유사한 결과로서, 단, dsDNA 도너의 경우에는 수백 개의 염기인데 반해, ssDNA 올리고의 경우에는 수십 bp 만큼의 고도로 압축된 거리 규모일 때이다.
(c) 일련의 돌연변이를 함유하는 단일 ssODN을 이용하고, 인접한 일련의 도입을 측정하는 유전자 전환 트랙에 대한 검정법. 중앙의 2 bp 미스매치 양측에 10 nt 만큼의 간격을 두고 이격되어 있는 3쌍의 2 bp 미스매치 (오렌지색)를 포함하는 ssODN 도너 (상단)를 사용하였다. 상기 영역을 서열분석하였을 때 >300,000개의 리드 중 ssODN에 의해 정의되는 미스매치를 >=1 보유하는 게놈 서열분석 리드는 거의 검출되지 않았다 (참고 문헌 62 (상기 문헌은 그 전문이 본원에서 참조로 포함된다) 참조). 상기 리드 모두 플롯팅하였고 (하단), 리드 서열은 색깔로 코딩되어 있다. 오렌지색: 정의된 미스매치; 녹색: 야생형 서열. 상기 ssODN을 사용하여 게놈 편집을 수행하였을 때, 오직 한가운데 돌연변이만이 도입된 패턴을 상기 경우의 85% (53/62)가 되게 하였고, 다른 경우에는 다중 B 미스매치가 도입되었다. 비록 B 도입 이벤트 횟수가 너무 낮아 길이가 > 10 bp인 트랙 분포를 추정하기 어렵기는 했지만, -10-10 bp의 짧은 트랙 영역이 지배적이라는 것은 명백하다.
도 12. Cas9-gRNA 뉴클레아제 및 닉카제 게놈 편집 효율.
뉴클레아제 (C2) (Cas9-gRNA) 또는 닉카제 (Cc) (Cas9D10A-gRNA) 및 상이한 배향 (Oc 및 On)의 ssODN의 조합으로 PGP1 iPSC를 공동 형질감염시켰다. 모든 ssODN은 그의 서열 한가운데에 게놈 DNA에 대하여 동일한 2 bp 미스매치를 포함하하였다. HDR 평가는 본원에 기술되어 있다.
도 13. 리TALE 서열 디자인 및 최적화
반복부를 제거하기 위해 수회의 디자인 사이클을 거쳐 리TALE 서열을 진화시켰다. 매회 사이클에서, 각 반복부로부터의 동의적 서열을 평가한다. 진화 DNA까지의 해밍 거리가 가장 큰 것을 선별한다. 최종 서열의 cai = 0.59이고, ΔG= -9.8 kcal/mol이다. 합성 단백질 디자인을 위해 상기의 일반 골격을 수행하기 위하여 R 패키지를 제공하였다.
도 14는 PGP1 세포에서의 Cas 9의 게놈 삽입의 PCR 확인을 보여주는 겔 영상이다. 라인 3, 6, 9, 12는 단순 PGP1 세포주의 PCR 생성물이다.
도 15는 유도하의 Cas9 mRNA의 mRNA 발현 수준을 나타낸 그래프이다.
도 16은 상이한 RNA 디자인에 의한 게놈 표적화 효율을 보여주는 그래프이다.
도 17은 가이드 RNA - 도너 DNA 융합체에 의해 달성된 44%의 상동성 재조합의 게놈 표적화 효율을 보여주는 그래프이다.
도 18은 본원에 기술된 시스템에 의해 생성된 동질유전자 PGP1 세포주의 유전자형을 보여주는 다이어그램이다. PGP1-iPS-BTHH는 BTHH 환자와 같은 단일 뉴클레오티드 결실 표현형을 가진다. PGP1-NHEJ는 다른 방식으로 프레임 쉬프트 돌연변이를 생성한 4 bp 결실을 가진다.
도 19는 환자 특이 세포에서 입증된 바, 동질유전자 PGP1 iPS로부터 유래된 심장 근육 세포가 결손 ATP 생산 및 F1F0 ATPase 특이 활성을 반복하였음을 보여주는 그래프이다.
도 1은 인간 체세포 및 줄기 세포에서의 리TALEN의 기능적 시험에 관한 것이다.
(a) 게놈 표적화 효율을 시험하기 위한 실험 디자인의 도식적 대표도. 종결 코돈 및 AAVS1 유전자좌로부터 유래된 68 bp 게놈 단편의 삽입에 의해 게놈적으로 통합된 GFP 코딩 서열을 파괴한다 (하단). tGFP 도너와의 뉴클레아제-매개 상동성 재조합에 의한 GFP 서열의 복원 (상단)을 통해 FACS에 의해 정량화될 수 있는 GFP+ 세포를 수득한다. 리TALEN 및 TALEN은 AAVS1 단편 내의 동일한 서열을 표적화한다.
(b) FACS에 의해 측정된, tGFP 도너만이 단독으로, tGFP 도너와 함께 TALEN, 및 tGFP 도너와 함께 리TALEN이 표적 유전자좌에 도입된 GFP+ 세포의 비율(%)을 도시한 막대 그래프. (N=3, 오차 막대=SD). 대표 FACS 플롯이 하기에 제시되어 있다.
(c) 천연 AAVS1 유전자좌에 대한 표적화 전략을 도시한 도식적 개요. 스플라이싱 억셉터 (SA)- 2A (자가-절단 펩티드), 퓨로마이신 저항성 유전자 (PURO) 및 GFP를 함유하는 도너 플라스미드가 기술되었다 (그 전문이 본원에 참조로 포함된 참고 문헌 10을 참조할 수 있다). 성공적인 편집 이벤트를 검출하는 데 사용된 PCR 프라이머의 위치는 청색 화살표로 표시되어 있다.
(d) 2주 동안 퓨로마이신 (0.5ug/mL)을 이용하여 PGP1 hiPSC의 성공적으로 표적화된 클론을 선별하였다. 대표적인 GFP+ 클론 3개에 대한 현미경 영상이 제시되어 있다. 세포를 또한 다능성 마커 TRA-1-60에 대하여 염색하였다. 스케일 바: 200 ㎛.
(e) 상기의 단일클론 GFP+ hiPSC 클론에서 수행된 PCR 검정법을 통해 도너 카세트가 AAVS1 부위에 성공적으로 삽입된 반면 (레인 1,2,3), 단순 hiPSC는 성공적인 삽입에 관한 어떤 증거도 보이지 않았다는 것 (레인 C)이 입증되었다.
도 2는 iPSC 중 CCR5 상에서의 리TALEN 및 Cas9-gRNA 게놈 표적화 효율 비교에 관한 것이다.
(a) 게놈 조작 실험 디자인의 도식적 대표도. 리TALEN 쌍 또는 Cas9-gRNA 표적화 부위에서 게놈 DNA에 대한 2 bp 미스매치를 보유하는 90mer ssODN을 리TALEN 또는 Cas9-gRNA 구축물과 함께 PGP1 hiPSC 내로 전달하였다. 도면에서 뉴클레아제의 절단 부위는 적색 화살표로 표시되어 있다.
(b) 리TALEN 쌍 (CCR5 #3) 및 ssODN, 또는 Cas9-gRNA 및 ssODN에 대한 HDR 및 NHEJ 효율에 관한 심층 서열분석 분석. hiPSC의 게놈 중의 변경을 GEAS에 의한 고처리량 서열 데이터로부터 분석하였다. 상단: HDR은 ssODN의 중앙부 내로 구축된 2 bp 점 돌연변이를 함유하는 리드의 분율로부터 정량화하고 (청색), NHEJ 활성은 게놈 내의 각 특이 위치에서의 결실 (회색)/삽입 (적색)의 분율로부터 정량화하였다. 리TALEN 및 ssODN 그래프의 경우, 녹색 점선을 플롯팅하여 두 리TALEN 결합 부위의 중앙부에 대해 상대적인 위치로 -26 bp 내지 +26 bp 위치인 리TALEN 쌍의 결합 부위의 외곽 경계를 마킹하였다. Cas9-gRNA 및 ssODN 그래프의 경우, 녹색 점선은 PAM 서열에 대해 상대적인 위치로 -20 내지 -1 bp 위치인 gRNA 표적화 부위의 외곽 경계를 마킹한다. 하단: 상기 명시된 바와 같이 처리된 전체 NHEJ 집단으로부터 분석된 hiPSC 중의 결실/삽입 크기 분포.
(c) PGP1 hiPSC에서의 리TALEN 및 Cas9-gRNA 표적화 CCR5의 게놈 편집 효율.
상단: CCR5에서 표적화된 게놈 편집 부위에 관한 도식적 대표도. 15개의 표적화 부위가 하기에 청색 화살표로 도시되어 있다. 각 부위에 대하여 세포를 리TALEN, 및 게놈 DNA에 대한 2 bp 미스매치를 보유하는 그의 상응하는 ssODN 도너로 이루어진 쌍으로 공동 형질감염시켰다. 형질감염 후 6일 경과하였을 때 게놈 편집 효율을 검정하였다. 유사하게, 15개의 Cas9-gRNA를 그의 상응하는 ssODN을 이용하여 개별적으로 PGP1-hiPSC 내로 형질감염시켜 같은 15개의 부위를 표적화하고, 형질감염 후 6일 경과하였을 때 효율을 분석하였다. 하단: PGP1 hiPSC에서의 리TALEN 및 Cas9-gRNA 표적화 CCR5의 게놈 편집 효율. 패널 1 및 2는 리TALEN에 의해 매개된 NHEJ 및 HDR 효율을 나타낸다. 패널 3 및 4는 Cas9-gRNA에 의해 매개된 NHEJ 및 HDR 효율을 나타낸다. NHEJ 비율은 표적화 부위에 결실 또는 삽입을 보유하는 게놈 대립 유전자의 빈도에 의해 계산하였고; HDR 비율은 2 bp 미스매치를 보유하는 게놈 대립 유전자의 빈도에 의해 계산하였다. 패널 5는 엔코드(ENCODE) 데이터베이스로부터의 hiPSC 세포주의 DNaseI HS 프로파일이다 (듀크(Duke) DNase HS, iPS NIHi7 DS). 특히, 다른 패널의 스케일은 상이하다.
(f) 3개의 표적화된 hiPSC 콜로니로부터의 PCR 앰플리콘의 생어(Sanger) 서열분석을 통해 게놈-삽입 경계에 예상 DNA 염기가 존재한다는 것을 확인하였다. M: DNA 래더. C: 단순 hiPSC 게놈 DNA를 포함하는 대조군.
도 3은 PGP1 hiPSC에서의 리TALEN 또는 Cas9-gRNA를 이용하는 ssODN-매개 HDR을 지배하는 기능적 파라미터에 관한 연구에 관한 것이다.
(a) PGP1 hiPSC를 리TALEN 쌍 (#3) 및 상이한 길이 (50, 70, 90, 110, 130, 150, 170 nts)의 ssODN으로 공동 형질감염시켰다. 모든 ssODN은 그의 서열 중간에 게놈 DNA에 대하여 동일한 2 bp 미스매치를 포함하였다. 90mer ssODN은 표적화된 게놈에서 최적의 HDR을 달성하였다. HDR, NHEJ 발생된 결실 및 삽입 효율 평가는 본원에 기술되어 있다.
(b) 각각이 중앙에 2 bp 미스매치 (A) 및 A로부터의 상이한 위치 상쇄부 (여기서, 상쇄부는 -30 bp→30 bp로 달랐다)에 추가의 2 bp 미스매치 (B)를 함유하는 리TALEN 쌍 #3에 상응하는 90mer ssODN을 사용하여 ssODN에 따른 상동성으로부터의 이탈의 효과를 시험하였다. 각 ssODN의 게놈 편집 효율은 PGP1 hiPSC에서 평가하였다. 하단의 막대 그래프는 표적화된 게놈에서의 오직 A만, 오직 B만, 및 A + B의 도입 빈도를 보여주는 것이다. HDR 비율은 중앙으로부터의 상동성 이탈 거리가 증가할수록 감소한다.
(c) 리TALEN 쌍 #3의 표적 부위로부터 다양한 거리 (-620 bp ~ 480 bp)를 두고 떨어져 있는 부위로 표적화되는 ssODN을 시험함으로써 돌연변이를 도입하는 데 ssODN이 배치될 수 있는 최대 거리를 평가하였다. 모든 ssODN은 그의 서열 한가운데에 2 bp 미스매치를 보유하였다. ssODN 미스매치가 리TALEN 쌍의 결합 부위의 한가운데로부터 40 bp만큼 떨어져 위치할 때 최소 HDR 효율 (<=0.06%)이 관찰되었다.
(d) PGP1 hiPSC를 Cas9-gRNA (AAVS1) 및 상이한 배향 (Oc: gRNA에 대해 상보적; On: gRNA에 대해 비상보적) 및 상이한 길이 (30, 50, 70, 90, 110 nt)의 ssODN으로 공동 형질감염시켰다. 모든 ssODN은 그의 서열 한가운데에 게놈 DNA에 대하여 동일한 2 bp 미스매치를 포함하였다. 70 mer Oc가 표적화된 게놈에서 최적의 HDR을 달성하였다.
도 4는 선별 없이 단일클론 게놈 편집된 hiPSC를 수득하기 위해 리TALEN 및 ssODN을 사용하는 것에 관한 것이다.
(a) 실험 타임라인.
(b) 도 2b에 기술된 NGS 플랫폼에 의해 평가된 리TALEN 쌍 및 ssODN (#3)의 게놈 조작 효율.
(c) 게놈 편집 이후의 단일클론 hiPSC 콜로니의 생어 서열분석 결과. 2 bp 이형접합성 유전자형 (CT/CT→TA/CT)이 PGP1-iPS-3-11, PGP1-iPS-3-13 콜로니의 게놈 내로 성공적으로 도입되었다.
(d) 표적화된 PGP1-iPS-3-11의 면역형광 염색. 세포를 다능성 마커 Tra-1-60 및 SSEA4에 대해 염색하였다.
(e) 단일클론 PGP1-iPS-3-11 세포로부터 생성된 테라토마 섹션의 헤마톡실린 및 에오신 염색.
도 5. 리TALE 디자인. (a) 리TALE-16.5에서의 단량체와 함께 원래의 TALE RVD 단량체로 이루어진 서열 정렬 (리TALE-M1→리TALE-M17). 원래의 서열로부터의 뉴클레오티드 변경은 회색으로 강조 표시되어 있다. (b) PCR에 의한 리TALE의 반복성 시험. 상단의 패널은 PCR 반응에서의 프라이머의 위치 및 리TALE/TALE의 구조를 도시한 것이다. 하단의 패널은 PCR 밴드를 도시한 것이고, 조건은 하기에 명시되어 있다. 원래의 TALE 주형 (우측 레인)과 함께 PCR 래더링이 제시되어 있다.
도 6. TALE 단일-인큐베이션 조립 (TASA) 조립 디자인 및 실시.
(a) TASA 조립을 위한 리TALE 이량체 블록의 라이브러리의 도식적 대표도. 2개의 RVD를 코딩하는 10개의 리TALE 이량체 블록의 라이브러리가 존재한다. 각 블록 내에서 16개의 이량체는 모두 RVD를 코딩하는 서열을 제외하면 동일한 DNA 서열을 공유한다; 다른 블록 중의 이량체는 독특한 서열을 가지지만, 그가 인접 블록과 32 bp의 중첩부를 공유하도록 디자인된다. 한 이량체 (블록6_AC)의 DNA 및 아미노산 서열은 우측에 열거되어 있다.
(b) TASA 조립의 도식적 대표도. 좌측 패널은 TASA 조립 방법을 도시한 것이다: 효소 혼합물/리TALE 블록/리TALE-N/TF 골격 벡터를 이용하여 원-포트 인큐베이션 반응을 수행한다. 반응 생성물은 박테리아 형질전환을 위해 직접 사용될 수 있다. 우측 패널은 TASA의 기전을 도시한 것이다. 37℃에서 엔도뉴클레아제에 의해 목표 벡터를 선형화하여 ccdB 역 선택 카세트를 절단하고; 블록의 단부 및 선형화된 벡터를 프로세싱하는 엑소뉴클레아제는 단편의 단부의 ssDNA 오버행을 노출시켜 블록 및 벡터 골격이 지정된 순서로 어닐링될 수 있도록 한다. 온도가 최대 50℃까지 상승하였을 때, 폴리머라제 및 리가제는 함께 작용하여 갭을 실링하고, 이로써, 형질전환을 위해 준비된 최종 구축물이 생성된다.
(c) 상이한 단량체 길이를 가지는 리TALE에 대한 TASA 조립 효율. 조립에 사용된 블록은 좌측에 도시되어 있고, 조립 효율은 우측에 제시되어 있다.
도 7. 렌티-리TALE의 기능성 및 서열 완전성.
도 8. GEAS의 감도 및 재현가능성.
(a) HDR 검출 한계의 정보 기반 분석. 리TALEN (#10)/ssODN인 데이터세트 전제하에, 예상되는 편집 (HDR)을 함유하는 리드를 확인하고, 이들 HDR 리드를 체계적으로 제거하여 편집 신호가 "희석된" 상이한 인공 데이터세트를 작성하였다. 제거율이 100, 99.8, 99.9, 98.9, 97.8, 89.2, 78.4, 64.9, 21.6, 10.8, 2.2, 1.1, 0.2, 0.1, 0.02, 및 0%인 HDR 리드를 포함하는 데이터세트는 HR 효율 범위가 0~0.67%인 인공 데이터세트로 작성되었다. 각각의 개별 데이터세트에 대하여, 배경 신호 (보라색), 및 표적화 부위에서 수득된 신호 (녹색)의 상호 정보 (MI)를 추정하였다. HDR 효율이 0.0014%를 초과할 때, 표적화 부위에서의 MI가 배경보다 현저히 더 높다. HDR 검출 한계는 0.0014% 내지 0.0071%인 것으로 추정되었다. MI 계산은 본원에 기술된 바와 같이 수행된다.
(b) 게놈 편집 평가 시스템의 재현가능성 시험. 플롯 쌍 (상단 및 하단)은 상기 명시된 리TALEN 쌍 및 세포 유형을 이용한 두 복제물의 HDR 및 NHEJ 평가 결과를 보여주는 것이다. 각 실험을 위해, 뉴클레오펙션, 표적화된 게놈 증폭, 심층 서열분석 및 데이터 분석을 독립적으로 수행하였다. 복제물의 게놈 편집 평가 편차는 √2(|HDR1-HDR2|)/((HDR+HDR2)/2) =ΔHDR/HDR 및 √2(|NHEJ1-NHEJ2|)/((NHEJ1+NHEJ2)/2) =ΔNHEJ/NHEJ로 계산하였고, 편차 결과는 하기 플롯에 열거되어 있다. 시스템의 평균 편차는 (19%+11%+4%+9%+10%+35%)/6=15%였다. 편차에 기여할 수 있는 인자로는 뉴클레오펙션하에 있는 세포의 상태, 뉴클레오펙션 효율, 및 서열분석 커버리지 및 품질을 포함한다.
도 9. CCR5 상에서의 리TALEN 및 Cas9-gRNA에 의한 NHEJ 및 HDR 효율의 통계하적 분석
(a) iPSC 중 동일 부위에서의 리TALEN에 의해 매개된 HR 및 NHEJ 효율의 상관관계 (r=0.91, P< 1X10-5).
(b) iPSC 중 동일 부위에서의 Cas9-gRNA에 의해 매개된 HR 및 NHEJ 효율의 상관관계 (r=0.74, P=0.002).
(c) iPSC에서의 Cas9-gRNA에 의해 매개된 NHEJ 효율 및 gRNA 표적화 부위의 Tm 온도의 상관관계 (r=0.52, P=0.04).
도 10. 게놈 편집 효율 및 후성적 상태의 상관관계 분석.
피어슨 상관관계를 사용하여 DNase I 감도와 게놈 조작 효율 (HR, NHEJ) 사이의 가능한 연관성을 연구하였다. 관찰된 상관관계를 임의 추출된 세트 (N=100000)와 비교하였다. 관찰된 상관관계가 모의 분포의 95번째 백분위수보다 더 높거나, 또는 5번째 백분위수보다 더 낮은 것은 잠재적으로 연관성이 있는 것으로 간주되었다. DNase I 감도와 NHEJ/HR 효율 사이에 어떤 뚜렷한 상관관계도 관찰되지 않았다.
도 11. ssODN-매개 게놈 편집에서의 상동성 쌍 형성의 영향.
(a) 도 3b에 기술된 실험에서, 중간 2 b 미스매치 (A)가 도입된 속도로 측정된 바, 전체 HDR은 제2 미스매치 B의 A로부터의 그의 거리가 증가함에 따라 감소하였다 (B에서 A까지의 상대적인 위치는 -30→30 bp로 다르다). B가 A로부터 단지 10 bp만큼 떨어져 있을 때 (-10 bp 내지 +10b)의 더 빠른 도입 속도는 dsDNA 절단에 인접한 게놈 DNA에 대한 ssODN의 쌍 형성이 덜 요구된다는 것을 반영하는 것일 수 있다.
(b) ssODN에 따른 유전자 전환 길이의 분포. A로부터의 B의 각 거리에서, HDR 이벤트 중 일부는 오직 A만을 도입하는 반면, 또 다른 일부는 A 및 B, 둘 모두를 도입한다. 상기 두 이벤트는 유전자 전환 트랙으로 해석될 수 있는데 (Elliott et al., 1998), 여기서, A+B 이벤트는 B 너머까지 확장된 긴 전환 트랙을 나타내고, 오직 A만의 이벤트는 B에 도달하지 못한 더 짧은 것을 나타낸다. 이러한 해석시, 올리고를 따른 양 방향의 유전자 전환 길이의 분포는 추정될 수 있다 (ssODN의 중간을 0으로, ssODN의 5' 단부쪽으로의 전환 트랙은 (-) 방향으로, 및 3' 단부 쪽으로의 것은 (+) 방향으로 정의된다). 유전자 전환 트랙은 그의 길이가 증가함에 따라 점진적으로 발생률이 감소하는데, 이 결과는 dsDNA 도너를 이용하였을 때 관찰된 유전자 전환 트랙 분포와 매우 유사한 결과로서, 단, dsDNA 도너의 경우에는 수백 개의 염기인데 반해, ssDNA 올리고의 경우에는 수십 bp 만큼의 고도로 압축된 거리 규모일 때이다.
(c) 일련의 돌연변이를 함유하는 단일 ssODN을 이용하고, 인접한 일련의 도입을 측정하는 유전자 전환 트랙에 대한 검정법. 중앙의 2 bp 미스매치 양측에 10 nt 만큼의 간격을 두고 이격되어 있는 3쌍의 2 bp 미스매치 (오렌지색)를 포함하는 ssODN 도너 (상단)를 사용하였다. 상기 영역을 서열분석하였을 때 >300,000개의 리드 중 ssODN에 의해 정의되는 미스매치를 >=1 보유하는 게놈 서열분석 리드는 거의 검출되지 않았다 (참고 문헌 62 (상기 문헌은 그 전문이 본원에서 참조로 포함된다) 참조). 상기 리드 모두 플롯팅하였고 (하단), 리드 서열은 색깔로 코딩되어 있다. 오렌지색: 정의된 미스매치; 녹색: 야생형 서열. 상기 ssODN을 사용하여 게놈 편집을 수행하였을 때, 오직 한가운데 돌연변이만이 도입된 패턴을 상기 경우의 85% (53/62)가 되게 하였고, 다른 경우에는 다중 B 미스매치가 도입되었다. 비록 B 도입 이벤트 횟수가 너무 낮아 길이가 > 10 bp인 트랙 분포를 추정하기 어렵기는 했지만, -10-10 bp의 짧은 트랙 영역이 지배적이라는 것은 명백하다.
도 12. Cas9-gRNA 뉴클레아제 및 닉카제 게놈 편집 효율.
뉴클레아제 (C2) (Cas9-gRNA) 또는 닉카제 (Cc) (Cas9D10A-gRNA) 및 상이한 배향 (Oc 및 On)의 ssODN의 조합으로 PGP1 iPSC를 공동 형질감염시켰다. 모든 ssODN은 그의 서열 한가운데에 게놈 DNA에 대하여 동일한 2 bp 미스매치를 포함하하였다. HDR 평가는 본원에 기술되어 있다.
도 13. 리TALE 서열 디자인 및 최적화
반복부를 제거하기 위해 수회의 디자인 사이클을 거쳐 리TALE 서열을 진화시켰다. 매회 사이클에서, 각 반복부로부터의 동의적 서열을 평가한다. 진화 DNA까지의 해밍 거리가 가장 큰 것을 선별한다. 최종 서열의 cai = 0.59이고, ΔG= -9.8 kcal/mol이다. 합성 단백질 디자인을 위해 상기의 일반 골격을 수행하기 위하여 R 패키지를 제공하였다.
도 14는 PGP1 세포에서의 Cas 9의 게놈 삽입의 PCR 확인을 보여주는 겔 영상이다. 라인 3, 6, 9, 12는 단순 PGP1 세포주의 PCR 생성물이다.
도 15는 유도하의 Cas9 mRNA의 mRNA 발현 수준을 나타낸 그래프이다.
도 16은 상이한 RNA 디자인에 의한 게놈 표적화 효율을 보여주는 그래프이다.
도 17은 가이드 RNA - 도너 DNA 융합체에 의해 달성된 44%의 상동성 재조합의 게놈 표적화 효율을 보여주는 그래프이다.
도 18은 본원에 기술된 시스템에 의해 생성된 동질유전자 PGP1 세포주의 유전자형을 보여주는 다이어그램이다. PGP1-iPS-BTHH는 BTHH 환자와 같은 단일 뉴클레오티드 결실 표현형을 가진다. PGP1-NHEJ는 다른 방식으로 프레임 쉬프트 돌연변이를 생성한 4 bp 결실을 가진다.
도 19는 환자 특이 세포에서 입증된 바, 동질유전자 PGP1 iPS로부터 유래된 심장 근육 세포가 결손 ATP 생산 및 F1F0 ATPase 특이 활성을 반복하였음을 보여주는 그래프이다.
본 발명의 측면은 예를 들어, 이중 가닥 핵산 절단에 의해 이루어지는 핵산 조작을 위한, 특정 반복부 서열이 결핍된 TALEN의 용도에 관한 것이다. 이중 가닥 핵산을 절단하는 데 TALEN을 사용하면, 비상동성 단부 연결 (NHEJ) 또는 상동성 재조합 (HR)이 이루어질 수 있다. 본 개시내용의 측면은 또한 예를 들어, 도너 핵산 존재하의 이중 가닥 핵산 절단에 의해 이루어지는 핵산 조작을 위한, 및 예컨대, 비상동성 단부 연결 (NHEJ) 또는 상동성 재조합 (HR)에 의한 도너 핵산의 이중 가닥 핵산 내로의 삽입을 위한, 특정 반복부 서열이 결핍된 TALEN의 용도도 고려한다.
전사 활성인자-유사 효과기 뉴클레아제 (TALEN)는 관련 기술분야에 공지되어 있고, TAL 효과기 DNA 결합 도메인을 DNA 절단 도메인에 융합시킴으로써 생성된 인공 제한 효소를 포함한다. 제한 효소는 특이 서열에서 DNA 가닥을 절단하는 효소이다. 전사 활성인자-유사 효과기 (TALE) 뉴클레아제는 원하는 DNA 서열에 결합하도록 조작될 수 있다. 문헌 [Boch, Jens (February 2011). "TALEs of genome targeting". Nature Biotechnology 29 (2): 135-6] (상기 문헌은 그 전문이 본원에서 참조로 포함된다)을 참조할 수 있다. 상기 조작된 TALE를 (DNA 가닥을 절단하는) DNA 절단 도메인과 조합함으로써 임의의 원하는 DNA 서열에 대해 특이적인 제한 효소인 TALEN이 제조된다. 특정 측면에 따라, TALEN을 계내 표적 핵산 편집을 위해, 예컨대, 계내 게놈 편집을 위해 세포 내로 도입한다.
한 측면에 따라, FokI 엔도뉴클레아제의 단부로부터의 비-특이 DNA 절단 도메인을 이용하여 효모 세포, 식물 세포 및 동물 세포에서 활성인 하이브리드 뉴클레아제를 구축할 수 있다. FokI 도메인은 적절한 배향 및 간격을 두고 표적 게놈 중의 부위에 대해 독특한 DNA 결합 도메인을 가진 두 구축물을 필요로 하면서, 이량체로서의 기능을 한다. TALE DNA 결합 도메인과 FokI 절단 도메인 사이의 아미노산 잔기의 개수, 및 두 개별 TALEN 결합 부위 사이의 염기의 개수, 둘 모두 활성에 영향을 미친다.
아미노산 서열과 TALE 결합 도메인의 DNA 인식 사이의 관계를 통해 디자인가능한 단백질을 얻을 수 있다. 소프트웨어 프로그램, 예컨대, DNA웍스(DNAWorks)를 이용하여 TALE 구축물을 디자인할 수 있다. TALE 구축물을 디자인하는 다른 방법은 통상의 기술자에게 공지되어 있다. 문헌 [Cermak, T.; Doyle, E. L.; Christian, M.; Wang, L.; Zhang, Y.; Schmidt, C.; Baller, J. A.; Somia, N. V. et al. (2011). "Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting". Nucleic Acids Research. doi:10.1093/nar/gkr218]; [Zhang, Feng; et.al. (February 2011). "Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription", Nature Biotechnology 29 (2): 149-53]; [Morbitzer, R.; Elsaesser, J.; Hausner, J.; Lahaye, T. (2011). "Assembly of custom TALE-type DNA binding domains by modular cloning". Nucleic Acids Research. doi:10'1093/nar/gkr151]; [Li, T.; Huang, S.; Zhao, X.; Wright, D. A.; Carpenter, S.; Spalding, M. H.; Weeks, D. P.; Yang, B. (2011). "Modularly assembled designer TAL effector nucleases for targeted gene knockout and gene replacement in eukaryotes". Nucleic Acids Research. doi"10.1093/nar/gkr188]; [Geiβler, R.; Scholze, H.; Hahn, S.; Streubel, J.; Bonas, U.; Behrens, S. E.; Boch, J. (2011). "Transcriptional Activators of Human Genes with Programmable DNA-Specificity". In Shiu, Shin-Han. PLoS ONE 6 (5): e19509]; [Weber, E.; Gruetzner, R.; Werner, S.; Engler, C.; Marillonnet, S. (2011). "Assembly of Designer TAL Effectors by Golden Gate Cloning". In Bendahmane, Mohammed. PLoS ONE 6 (5): e19722] (상기 문헌들은 그 전문이 본원에서 참조로 포함된다)을 참조할 수 있다.
예시적인 측면에 따라, 일단 TALEN 유전자를 조립하고 나면, 이를 특정 실시양태에 따라 플라스미드로 내로 삽입할 수 있고; 이어서, 플라스미드를 이용하여 표적 세포를 형질감염시키고, 상기 표적 세포에서 유전자 생성물이 발현되고, 이는 핵으로 진입하여 게놈에 접근하게 된다. 예시적인 측면에 따라, 본원에 기술된 바와 같은 TALEN을 이용하여, 세포는 수복 기전으로 대응하는 이중 가닥 절단 (DSB)을 유도함으로써 표적 핵산, 예컨대, 게놈을 편집할 수 있다. 예시적인 수복 기전으로는 어닐링을 위한 서열 중복부가 거의 없거나, 전혀 없는 이중 가닥 절단의 양측으로부터의 DNA를 재연결합하는 비-상동성 단부 연결 (NHEJ)을 포함한다. 이러한 수복 기전은 삽입 또는 결실 (indel), 또는 염색체 재배열을 통하여 게놈 중 오류를 유도하고; 상기와 같은 임의의 오류로 인하여 상기 위치에서 코딩된 유전자 생성물은 비-기능성이 될 수 있다. 문헌 [Miller, Jeffrey; et.al. (February 2011). "A TALE nuclease architecture for efficient genome editing". Nature Biotechnology 29 (2): 143-8] (상기 문헌은 그 전문이 본원에서 참조로 포함된다)을 참조할 수 있다. 이러한 활성은 사용되는 종, 세포 유형, 표적 유전자, 및 뉴클레아제에 따라 달라질 수 있기 때문에, PCR에 의해 증폭되는 두 대립유전자 사이의 임의의 차이를 검출하는 이종이중체 절단 검정법을 이용함으로써 활성을 모니터링할 수 있다. 절단 생성물은 단순 아가로스 겔 또는 슬랩 겔 시스템 상에서 시각화될 수 있다.
별법으로, DNA는 외인성 이중 가닥 DNA 단편의 존재하에서 NHEJ를 통해 게놈 내로 도입될 수 있다. 형질감염된 이중 가닥 서열이 수복 효소에 대한 주형으로서 사용되는 바, 상동성 지정 수복 또한 DSB에 외래 DNA를 도입할 수 있다. 특정 측면에 따라, 본원에 기술된 TALEN을 이용하여 안정적으로 변형된 인간 배아 줄기 세포 및 유도 다능성 줄기 세포 (IPSC) 클론을 생성할 수 있다. 특정 측면에 따라, 본원에 기술된 TALEN을 이용하여 녹아웃 종, 예컨대, C. 엘레강스(C. elegans), 녹아웃 래트, 녹아웃 마우스 또는 녹아웃 제브라피시를 생성할 수 있다.
본 개시내용의 한 측면에 따라, 실시양태는 줄기 세포 내에서 DNA로 공동 국재화하고, DNA를 분해 또는 절단시키면서, 외인성 DNA를 삽입하기 위한, 외인성 DNA, 뉴클레아제 효소, 예컨대, DNA 결합 단백질 및 가이드 RNA의 용도에 관한 것이다. 상기 DNA 결합 단백질은 다양한 목적을 위해 DNA에 결합하는 것으로 통상의 기술자에게 쉽게 공지되어 있다. 상기 DNA 결합 단백질은 천연적으로 발생된 것일 수 있다. 본 개시내용의 범주 내에 포함되는 DNA 결합 단백질로는, 본원에서 가이드 RNA로 지칭되는 RNA에 의해 가이드될 수 있는 것을 포함한다. 상기 측면에 따라, 가이드 RNA 및 RNA에 의해 가이드되는 DNA 결합 단백질은 DNA에서 공동 국재화 복합체를 형성한다. 뉴클레아제 활성을 가진 상기 DNA 결합 단백질은 통상의 기술자에게 공지되어 있고, 이는 뉴클레아제 활성을 가지는 천연적으로 발생된 DNA 결합 단백질, 예를 들어, II형 CRISPR 시스템에 존재하는 Cas9 단백질과 같은 것을 포함한다. 상기 Cas9 단백질 및 II형 CRISPR 시스템은 관련 기술분야에 문서로 잘 기록되어 있다. 문헌 [Makarova et al., Nature Reviews, Microbiology, Vol. 9, June 2011, pp. 467-477] (모든 보충 정보 포함) (상기 문헌은 그 전문이 본원에서 참조로 포함된다)을 참조할 수 있다.
뉴클레아제 활성을 가진 예시적인 DNA 결합 단백질은 이중 가닥 DNA를 니킹하거나, 또는 절단하는 기능을 한다. 상기 뉴클레아제 활성은 뉴클레아제 활성을 보이는 하나 이상의 폴리펩티드 서열을 가진 DNA 결합 단백질로부터 생성될 수 있다. 상기 예시적인 DNA 결합 단백질은, 각 도메인이 이중 가닥 DNA의 특정 가닥을 절단 또는 니킹하는 것을 담당하는 것인, 별개의 두 뉴클레아제 도메인을 가질 수 있다. 통상의 기술자에게 공지된 뉴클레아제 활성을 가진 예시적인 폴리펩티드 서열은 McrA-HNH 뉴클레아제 관련 도메인 및 RuvC-유사 뉴클레아제 도메인을 포함한다. 따라서, 예시적인 DNA 결합 단백질은 자연상에서 McrA-HNH 뉴클레아제 관련 도메인 및 RuvC-유사 뉴클레아제 도메인 중 하나 이상의 것을 함유하는 것이다.
예시적인 DNA 결합 단백질은 II형 CRISPR 시스템의 RNA에 의해 가이드되는 DNA 결합 단백질이다. 예시적인 DNA 결합 단백질은 Cas9 단백질이다.
S. 피오게네스에서, Cas9는 단백질 중 두 촉매성 도메인: DNA의 상보적인 가닥을 절단하는 HNH 도메인 및 비-상보적인 가닥을 절단하는 RuvC-유사 도메인에 이해 매개되는 프로세스를 통해 프로토스페이서-인접 모티프 (PAM)로부터 3 bp만큼 상류쪽에 평활 말단 이중 가닥 절단을 생성한다. 문헌 [Jinke et al., Science 337, 816-821 (2012)] (상기 문헌은 그 전문이 본원에서 참조로 포함된다)를 참조할 수 있다. Cas9 단백질은 문헌 [Makarova et al., Nature Reviews, Microbiology, Vol. 9, June 2011, pp. 467-477]에 대한 보충 정보에서 확인되는 것과 같은 하기의 것을 비롯한 다수의 II형 CRISPR 시스템: 메타노코쿠스 마리팔루디스(Methanococcus maripaludis) C7; 코리네박테리움 디프테리아(Corynebacterium diphtheriae); 코리네박테리움 에피시엔스(Corynebacterium efficiens) YS-314; 코리네박테리움 글루타미쿰(Corynebacterium glutamicum) ATCC 13032 키타사토(Kitasato); 코리네박테리움 글루타미쿰 ATCC 13032 빌레펠트(Bielefeld); 코리네박테리움 글루타미쿰 R; 코리네박테리움 크롭펜스테드티이(Corynebacterium kroppenstedtii) DSM 44385; 마이코박테리움 엡세수스(Mycobacterium abscessus) ATCC 19977; 노카르디아 파르시니카(Nocardia farcinica) IFM10152; 로도코쿠스 에리트로폴리스(Rhodococcus erythropolis) PR4; 로도코쿠스 조스티이(Rhodococcus jostii) RHA1; 로도코쿠스 오파쿠스(Rhodococcus opacus) B4 uid36573; 아시도써무스 셀룰롤리티쿠스(Acidothermus cellulolyticus) 11B; 아르트로박터 클로로페놀리쿠스(Arthrobacter chlorophenolicus) A6; 크립벨라 플라비다(Kribbella flavida) DSM 17836 uid43465; 써모모노스포라 쿠르바타(Thermomonospora curvata) DSM 43183; 비피도박테리움 덴티움(Bifidobacterium dentium) Bd1; 비피도박테리움 롱검(Bifidobacterium longum) DJO10A; 슬랙키아 헬리오트리니레두센스(Slackia heliotrinireducens) DSM 20476; 퍼셉포넬라 마리나(Persephonella marina) EX H1; 박테로이데스 프라질리스(Bacteroides fragilis) NCTC 9434; 카프노시토파가 오크라세아(Capnocytophaga ochracea) DSM 7271; 플라보박테리움 프시크로필룸(Flavobacterium psychrophilum) JIP02 86; 아커만시아 뮤시니필라(Akkermansia muciniphila) ATCC BAA 835; 로세이플렉수스 카스텐홀지이(Roseiflexus castenholzii) DSM 13941; 로세이플렉수스 RS1; 시네코시스티스(Synechocystis) PCC6803; 엘루시마이크로비움 미누툼(Elusimicrobium minutum) Pei191; 미배양된 흰개미(Termite) 군 1 박테리움 계통형 Rs D17; 피브로박터 숙시노게네스(Fibrobacter succinogenes) S85; 바실럿 세레우스(Bacillus cereus) ATCC 10987; 리스테리아 인노쿠아(Listeria innocua); 락토바실러스 카제이(Lactobacillus casei); 락토바실러스 람노수스(Lactobacillus rhamnosus) GG; 락토바실러스 살리바리우스(Lactobacillus salivarius) UCC118; 스트렙토코쿠스 아갈락티아에(Streptococcus agalactiae) A909; 스트렙토코쿠스 아갈락티아에 NEM316; 스트렙토코쿠스 아갈락티아에 2603; 스트렙토코쿠스 디스갈락티아에 이퀴시밀리스(Streptococcus dysgalactiae equisimilis) GGS 124; 스트렙토코쿠스 이퀴 주에피데미쿠스(Streptococcus equi zooepidemicus) MGCS10565; 스트렙토코쿠스 갈로리티쿠스(Streptococcus gallolyticus) UCN34 uid46061; 스트렙토코쿠스 고르도니이 칼리스 섭스트(Streptococcus gordonii Challis subst) CH1; 스트렙토코쿠스 뮤탄스(Streptococcus mutans) NN2025 uid46353; 스트렙토코쿠스 뮤탄스; 스트렙토코쿠스 피오게네스 M1 GAS; 스트렙토코쿠스 피오게네스 MGAS5005; 스트렙토코쿠스 피오게네스 MGAS2096; 스트렙토코쿠스 피오게네스 MGAS9429; 스트렙토코쿠스 피오게네스 MGAS10270; 스트렙토코쿠스 피오게네스 MGAS6180; 스트렙토코쿠스 피오게네스 MGAS315; 스트렙토코쿠스 피오게네스 SSI-1; 스트렙토코쿠스 피오게네스 MGAS10750; 스트렙토코쿠스 피오게네스 NZ131; 스트렙토코쿠스 써모필레스(Streptococcus thermophiles) CNRZ1066; 스트렙토코쿠스 써모필레스 LMD-9; 스트렙토코쿠스 써모필레스 LMG 18311; 클로스트리디움 보툴리눔(Clostridium botulinum) A3 로크 마리(Loch Maree); 클로스트리디움 보툴리눔 B 에클룬드(Eklund) 17B; 클로스트리디움 보툴리눔 Ba4 657; 클로스트리디움 보툴리눔 F 랑엘란드(Langeland); 클로스트리디움 셀룰로리티쿰(Clostridium cellulolyticum) H10; 피네골디아 마그나(Finegoldia magna) ATCC 29328; 유박테리움 렉탈레(Eubacterium rectale) ATCC 33656; 미코플라스마 갈리셉티쿰(Mycoplasma gallisepticum); 미코플라스마 모빌레(Mycoplasma mobile) 163K; 미코플라스마 페네트란스(Mycoplasma penetrans); 미코플라스마 시노비아에(Mycoplasma synoviae) 53; 스트렙토바실러스 모닐리포르미스(Streptobacillus moniliformis) DSM 12112; 브라디리조비움(Bradyrhizobium) BTAi1; 니트로박터 함부르겐시스(Nitrobacter hamburgensis) X14; 로도슈도모나스 팔루스트리스(Rhodopseudomonas palustris) BisB18; 로도슈도모나스 팔루스트리스 BisB5; 파르비바쿨럼 라바멘티보란스(Parvibaculum lavamentivorans) DS-1; 디노로세오박터 쉬바에(Dinoroseobacter shibae) DFL 12; 글루코나세토박터 디아조트로피쿠스(Gluconacetobacter diazotrophicus) Pal 5 FAPERJ; 글루코나세토박터 디아조트로피쿠스 Pal 5 JGI; 아조스피릴룸(Azospirillum) B510 uid46085; 로도스피릴룸 루브룸(Rhodospirillum rubrum) ATCC 11170; 디아포로박터(Diaphorobacter) TPSY uid29975; 베르미네프로박터 에이세니아에(Verminephrobacter eiseniae) EF01-2; 네이세리아 메닌기티데스(Neisseria meningitides) 053442; 네이세리아 메닌기티데스 알파14; 네이세리아 메닌기티데스 Z2491; 데술포비브리오 살렉시겐스(Desulfovibrio salexigens) DSM 2638; 캄필로박터 제주니 도일레이(Campylobacter jejuni doylei) 269 97; 캄필로박터 제주니 81116; 캄필로박터 제주니; 캄필로박터 라리(Campylobacter lari) RM2100; 헬리코박터 헤파티쿠스(Helicobacter hepaticus); 올리넬라 숙시노게네스(Wolinella succinogenes); 톨루모나스 아우엔시스(Tolumonas auensis) DSM 9187; 슈도알테로모나스 아틀란티카(Pseudoalteromonas atlantica) T6c; 쉬와넬라 페알레아나(Shewanella pealeana) ATCC 700345; 레지오넬라 뉴모필라(Legionella pneumophila) 파리스(Paris); 악티노바실러스 숙시노게네스(Actinobacillus succinogenes) 130Z; 파스퇴렐라 물토시다(Pasteurella multocida); 프란시셀라 투라렌시스 노비시다(Francisella tularensis novicida) U112; 프란시셀라 투라렌시스 홀라르티카(Francisella tularensis holarctica); 프란시셀라 투라렌시스 FSC 198; 프란시셀라 투라렌시스 투라렌시스(Francisella tularensis tularensis); 프란시셀라 투라렌시스 WY96-3418; 및 트레포네마 덴티콜라(Treponema denticola) ATCC 35405에 존재하는 것으로 공지되어 있다. 따라서, 본 개시내용의 측면은 II형 CRISPR 시스템에 존재하는 Cas9 단백질에 관한 것이다.
Cas9 단백질은 통상의 기술자에 의해 문헌에서 Csn1로 지칭될 수 있다. S. 피오게네스 Cas9 단백질은 하기에 제시되어 있다. 문헌 [Deltcheva et al., Nature 471, 602-607 (2011)] (상기 문헌은 그 전문이 본원에서 참조로 포함된다)을 참조할 수 있다.
한 측면에 따라, RNA에 의해 가이드되는 DNA 결합 단백질은 DNA에 결합할 수 있고, RNA에 의해 가이드될 수 있고, DNA를 절단할 수 있는 단백질의 능력을 유지하는 Cas9의 호모로그 및 오르토로그를 포함한다. 한 측면에 따라, Cas9 단백질은 S. 피오게네스로부터의 천연적으로 발생된 Cas9에 대해 기재된 것과 같은 서열, 및 그와 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95%, 98% 또는 99% 이상 상동성이고, DNA 결합 단백질, 예컨대, RNA에 의해 가이드되는 DNA 결합 단백질인 것인 단백질 서열을 포함한다.
한 측면에 따라, 줄기 세포에서 부위 특이적인 방식으로 RNA에 의해 가이드되는 게놈 절단을 수행할 수 있고, 원하는 경우, 외인성 도너 핵산의 삽입에 의해 줄기 세포 게놈을 변형시킬 수 있는 조작된 Cas9-gRNA 시스템을 제공한다. 가이드 RNA는 DNA 상의 표적 부위 또는 표적 유전자좌에 상보적이다. 가이드 RNA는 crRNA-tracrRNA 키메라일 수 있다. 가이드 RNA는 세포 주변의 배지로부터 도입될 수 있다. 이러한 방식으로 각종 가이드 RNA를 주변 배지에 제공하고, 세포에 의해 가이드 RNA가 흡수되고, 배지를 추가의 가이드 RNA로 보충하는 정도로 세포를 연속적으로 변형시키는 방법을 제공한다. 보충은 연속적인 방식으로 이루어질 수 있다. Cas9는 표적 게놈 DNA에 또는 그 주변에 결합한다. 하나 이상의 가이드 RNA는 표적 게놈 DNA에 또는 그 주변에 결합한다. Cas9는 표적 게놈 DNA를 절단하고, 외인성 도너 DNA가 DNA 내로 그 절단 부위에 삽입된다.
따라서, 본 방법은 RNA를 코딩하는 (또는 주변 배지로부터 RNA를 제공하는) 핵산 및 외인성 도너 핵산을 삽입하고, RNA를 발현하고 (또는 RNA를 흡수하고), DNA를 절단하는 방식으로 RNA, Cas9 및 DNA를 공동 국재화시키고, 외인성 도너 핵산을 삽입하는 것을 사이클링함으로써 Cas9를 발현하는 줄기 세포 내에서 외인성 도너 핵산을 DNA 내로 다중으로 삽입하기 위한 Cas9 단백질 및 외인성 도너 핵산과 함께 가이드 RNA의 용도에 관한 것이다. 본 방법 단계는 임의의 원하는 개수만큼 DNA를 변형시킬 수 있도록 임의의 원하는 횟수로 사이클링될 수 있다. 따라서, 본 개시내용의 방법은 본원에 개시된 Cas9 단백질 및 가이드 RNA를 사용하여 표적 유전자를 편집하여 줄기 세포를 다중으로 유전적 및 후성적으로 조작하는 것에 관한 것이다.
추가로, 본 개시내용의 측면은 일반적으로 줄기 세포, 예컨대, 인간 줄기 세포의 DNA, 예컨대, 게놈 DNA 내로의 외인성 도너 핵산의 다중 삽입을 위한 DNA 결합 단백질 또는 시스템 (예컨대, 본원에 기술된 변형된 TALENS 또는 Cas9)의 용도에 관한 것이다. 통상의 기술자는 본 개시내용에 기초하여 예시적인 DNA 결합 시스템을 쉽게 확인할 수 있을 것이다.
달리 명시되지 않는 한, 본 개시내용에 따른 세포로는 본원에 기술된 바와 같이 외래 핵산이 그 내부로 도입될 수 있고, 거기서 발현될 수 있는 임의의 세포를 포함한다. 본원에 기술된 본 개시내용의 기본 개념은 세포 유형에 의해 한정되지 않는다는 것을 이해하여야 한다. 본 개시내용에 따른 세포로는 체세포, 줄기 세포, 진핵 세포, 원핵 세포, 동물 세포, 식물 세포, 진균 세포, 고세균 세포, 진정세균 세포 등을 포함한다. 세포로는 진핵 세포 예컨대, 효모 세포, 식물 세포, 및 동물 세포를 포함한다. 특정 세포로는 포유동물 세포, 예컨대, 인간 세포를 포함한다. 추가로, 세포로는 DNA를 변형시키는 것이 유익하거나, 바람직한 임의의 것을 포함한다.
표적 핵산으로는, 본원에 기술된 바와 같은 뉴클레아제 활성을 가지는 TALEN 또는 RNA에 의해 가이드되는 DNA 결합 단백질이 그를 니킹 또는 절단하는 데 유용할 수 있는 것인 임의의 핵산 서열을 포함한다. 표적 핵산으로는 본원에 기술된 바와 같은 공동 국재화 복합체가 그를 니킹 또는 절단하는 데 유용할 수 있는 것인 임의의 핵산 서열을 포함한다. 표적 핵산으로는 유전자를 포함한다. 본 개시내용의 목적을 위해, DNA, 예컨대, 이중 가닥 DNA는 표적 핵산을 포함할 수 있고, 공동 국재화 복합체는 DNA에 결합할 수 있거나, 또는 다르게는 그와 공동 국재화할 수 있거나, 또는 TALEN는 다르게는 공동 국재화 복합체 또는 TALEN이 표적 핵산에 대하여 원하는 효과를 발휘할 수 있는 방식으로 표적 핵산에서 또는 그에 인접하게 또는 그 부근에서 DNA에 결합할 수 있다. 상기 표적 핵산은 내인성 (또는 천연적으로 발생된) 핵산 및 외인성 (또는 외래) 핵산을 포함할 수 있다. 본 개시내용에 기초하여 통상의 기술자는 DNA로 공동 국재화하는 가이드 RNA 및 Cas9 단백질, 또는 DNA에 결합하는 TALEN을 쉽게 확인하거나, 또는 디자인할 수 있을 것이다. 통상의 기술자는 추가로, 유사하게, 표적 핵산을 비롯한 DNA로 공동 국재화하는 전사 조절인자 단백질 또는 도메인, 예컨대, 전사 활성인자 또는 전사 억제인자를 확인할 수 있을 것이다. DNA로는 게놈 DNA, 미토콘드리아 DNA, 바이러스 DNA 또는 외인성 DNA를 포함한다. 한 측면에 따라, 본 개시내용의 실시에서 유용한 물질 및 방법으로는 예시적인 균주 및 배지, 플라스미드 구축, 플라스미드 형질전환, 일과성 gRNA 카세트 및 도너 핵산의 전기천공, 도너 DNA를 포함하는 gRNA 플라스미드의 Cas9-발현 세포로의 형질전환, Cas9의 갈락토스 유도, 효모 게놈에서의 CRISPR-Cas 표적 확인 등을 비롯한, 문헌 [Di Carlo, et al., Nucleic Acids Research, 2013, vol. 41, No. 7 4336-4343] (상기 문헌은 그 전문이 모든 목적을 위해 본원에서 참조로 포함된다)에 기술되어 있는 것을 포함한다. 본 발명을 수행하는 데 있어 통상의 기술자에게 유용한 정보, 물질 및 방법을 비롯한 추가의 참조 사항은 문헌 [Mali,P., Yang,L., Esvelt,K.M., Aach,J., Guell,M., DiCarlo,J.E., Norville,J.E. and Church,G.M. (2013) RNA-Guided human genome engineering via Cas9. Science, 10.1126fscience.1232033]; [Storici,F., Durham,C.L., Gordenin,D.A. and Resnick,M.A. (2003) Chromosomal site-specific double-strand breaks are efficiently targeted for repair by oligonucleotides in yeast. PNAS, 100, 14994-14999] 및 [Jinek,M., Chylinski,K., Fonfara,l., Hauer,M., Doudna,J.A. and Charpentier,E. (2012) A programmable dual-RNA-Guided DNA endonuclease in adaptive bacterial immunity. Science, 337, 816-821] (상기 문헌들은 각각 그 전문이 모든 목적을 위해 본원에서 참조로 포함된다)에 제공되어 있다.
외래 핵산 (즉, 세포의 천연 핵산 조성의 일부가 아닌 것)은 상기 도입을 위해 통상의 기술자에게 공지된 임의의 방법을 사용하여 세포 내로 도입될 수 있다. 상기 방법으로는 형질감염, 형질도입, 바이러스 형질도입, 미세주입, 리포펙션, 뉴클레오펙션, 나노입자 충격법, 형질전환, 접합 등을 포함한다. 통상의 기술자는 쉽게 확인가능한 문헌 자료를 이용하여 상기 방법을 쉽게 이해하고, 적합화시킬 것이다.
도너 핵산은 본원에 기술된 바와 같이 핵산 서열 내로 삽입되는 임의의 핵산을 포함한다.
하기 실시예는 본 개시내용을 대표하는 것으로서 기술되는 것이다. 상기 실시양태 및 다른 등가의 실시양태는 본 개시내용, 도면 및 첨부된 청구범위를 고려하면 자명한 바, 본 실시예는 본 개시내용의 범주를 한정하는 것으로 해석되지 않아야 한다.
실시예 I
가이드 RNA 조립
선택된 표적 서열의 19 bp (즉, 5'-N19-NGG-3'의 5'-N19)를 2개의 상보적인 100 mer 올리고뉴클레오티드 내로 도입하였다 (TTCTTGGCTTTATATATCTTGTGGAAAGGACGAAACACCGN19GTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTAGTCC). 각 100 mer 올리고뉴클레오티드를 물 중에 100 mM으로 현탁시키고, 동량으로 혼합하고, 써모사이클 장치 (95℃, 5 min; 4℃까지, 0.1℃/sec의 경사로)에서 어닐링시켰다. 목표 벡터를 제조하기 위해, AfIII를 이용하여 gRNA 클로닝 벡터 (애드진(Addgene) 플라스미드 ID 41824)를 선형화시키고, 벡터를 정제하였다. 10 ng 어닐링된 100 bp 단편, 100 ng 목표 골격, 1X 깁손(Gibson) 조립 반응 믹스 (뉴 잉글랜드 바이오랩스(New England Biolabs))를 이용하여 50℃에서 30 min 동안 (10 ul) gRNA 조립 반응을 수행하였다. 박테리아 형질전환을 위해서 직접 본 반응을 프로세싱하여 개별 조립체를 전이증식(colonize)시킬 수 있다.
실시예 II
재코딩된 TALE 디자인 및 조립
조립을 촉진시키고, 발현을 개선시키기 위해 리TALE를 다른 수준으로 최적화시켰다. 먼저 리TALE DNA 서열을 인간 코돈 선호도, 및 5' 단부의 낮은 mRNA 폴딩 에너지에 맞게 함께 최적화시켰다 (진GA(GeneGA), 바이오컨덕터(Bioconductor)). 수회의 사이클을 거쳐 수득된 서열을 진화시켜 11 bp 초과의 반복부 (동방향 또는 역방향)를 제거하였다 (도 12 참조). 각 사이클에서 각 반복부에 대한 동의적 서열을 평가하였다. 진화 DNA까지의 해밍 거리가 가장 큰 것을 선별하였다. 16.5개의 단량체를 가지는 리TALE 중 하나의 서열은 하기와 같다:
특정 측면에 따라, 상기 서열과 80% 이상의 서열 동일성, 85% 이상의 서열 동일성, 90% 이상의 서열 동일성, 95% 이상의 서열 동일성, 98% 이상의 서열 동일성, 또는 99% 이상의 서열 동일성을 가지는 TALE가 사용될 수 있다. TALE의 DNA 결합 활성은 그대로 유지하면서, 상기 서열은 달라질 수 있는 경우를 통상의 기술자는 쉽게 이해할 것이다.
표준 카파 HIFI (KPAP) PCR 조건하에, 1회차 PCR에서는 RVD 코딩 서열을 도입하였고, 2회차 PCR에서는 인접 블록과의 36 bp의 중복부를 포함하는 전체 이량체 블록을 생성한, 2회에 걸친 PCR에 의해 2개의 RVD를 코딩하는 리TALE 이량체 블록 (도 6a 참조)을 생성하였다. QIA퀵(QIAquick) 96 PCR 정제 키트 (퀴아젠(QIAGEN))를 이용하여 PCR 생성물을 정제하고, 나노-드롭(Nano-drop)에 의해 농도를 측정하였다. 프라이머 및 주형 서열은 하기 표 1 및 표 2에 열거되어 있다.
<표 1>
<표 2>
TALE-TF 및 TALEN 클로닝 골격을 변형시킴으로써 리TALEN 및 리TALE-TF 목표 벡터를 구축하였다 (참고 문헌 24 (상기 문헌은 그 전문이 본원에서 참조로 포함된다) 참조). 벡터 상의 0.5 RVD 영역을 재코딩하고, SapI 절단 부위를 지정된 리TALE 클로닝 부위에 도입하였다. 리TALEN 및 리TALE-TF 골격의 서열은 도 20에 제공되어 있다. 플라스미드를 제조사가 권고한 조건에 따라 SapI (뉴 잉글랜드 바이오랩스)로 미리 처리하고, QIA퀵 PCR 정제용 키트 (퀴아젠)로 정제할 수 있다.
200 ng의 각 블록, 500 ng 목표 골격, 1X TASA 효소 혼합물 (2 U SapI, 100 U 앰프리가제(Ampligase) (에피상트르(Epicentre)), 10 mU T5 엑소뉴클레아제 (에피상트르), 2.5 U 퓨전(Phusion) DNA 폴리머라제 (뉴 잉글랜드 바이오랩스)) 및 상기 기술된 바와 같은 1X 등온 조립 반응 완충제 (참고 문헌 25 (상기 문헌은 그 전문이 본원에서 참조로 포함된다) 참조) (5% PEG-8000, 100 mM 트리스-HCl (pH 7.5), 10 mM MgCl2, 10 mM DTT, 0.2 mM의 4가지의 dNTP 각각 및 1 mM NAD)를 이용하여 (10 ul) 원-포트 TASA 조립 반응을 수행하였다. 37℃에서 5 min 및 50℃에서 30 min 동안 인큐베이션을 수행하였다. 박테리아 형질전환을 위해 직접 TASA 조립 반응을 프로세싱하여 개별 조립체를 전이증식시킬 수 있다. 본 접근법을 이용한 경우, 전장의 구축물의 수득률은 ~20%이다. 별법으로, >90% 효율은 3단계 조립에 의해 달성될 수 있다. 먼저, 50℃에서 30 min 동안 200 ng의 각 블록, 1X 리TALE 효소 혼합물 (100 U 앰프리가제, 12.5 mU T5 엑소뉴클레아제, 2.5 U 퓨전 DNA 폴리머라제) 및 1X 등온 조립 완충제를 이용하여 10 ul 리TALE 조립 반응을 수행한 후, 표준화된 카파 HIFI PCR 반응, 아가로스 겔 전기영동, 및 QIA퀵 겔 추출 (퀴아젠(Qiagen))을 수행하여 전장의 리TALE를 농축시킨다. 이어서, 200 ng 리TALE 앰플리콘을 500 ng의, SapI로 미리 처리된 목표 골격, 1X 리TALE 조립 혼합물 및 1X 등온 조립 반응 완충제와 함께 혼합하고, 50 ℃에서 30 min 동안 인큐베이션시킬 수 있다. 박테리아 형질전환을 위해 직접 리TALE 최종 조립 반응을 프로세싱하여 개별 조립체를 전이증식시킬 수 있다. 통상의 기술자는 공지된 것으로부터 엔도뉴클레아제, 엑소뉴클레아제, 폴리머라제 및 리가제를 쉽게 선택함으로써 본원에 기술된 방법을 실시할 수 있다. 예를 들어, II형 엔도뉴클레아제, 예컨대, FokI, BtsI, EarI, SapI가 사용될 수 있다. 적정가능한 엑소뉴클레아제, 예컨대, 람다 엑소뉴클레아제, T5 엑소뉴클레아제 및 엑소뉴클레아제 III이 사용될 수 있다. 비-핫스타트(hotstart) 폴리머라제, 예컨대, 퓨전 DNA 폴리머라제, Taq DNA 폴리머라제 및 VentR DNA 폴리머라제가 사용될 수 있다. 열안정한 리가제, 예컨대, 앰프리가제, pfu DNA 리가제, Taq DNA 리가제가 본 반응에서 사용될 수 있다. 추가로, 사용되는 특정 종에 따라 상기 엔도뉴클레아제, 엑소뉴클레아제, 폴리머라제 및 리가제를 활성화시키는 데 상이한 반응 조건이 사용될 수 있다.
실시예 III
세포주 및 세포 배양
PGP1 iPS 세포를 mTeSR1 (스템셀 테크놀러지즈 Technologies(Stemcell Technologies)) 중 마트리겔 (BD 바이오사이언시즈(BD Biosciences))-코팅 플레이트 상에 유지시켰다. 배양물을 매 5-7일마다 TrypLE 익스프레스(TrypLE Express) (인비트로겐(Invitrogen))로 계대접종하였다. 293T 및 293FT 세포를 10% 우태아 혈청 (FBS, 인비트로겐), 페니실린/스트렙토마이신 (pen/strep, 인비트로겐), 및 비필수 아미노산 (NEAA, 인비트로겐)으로 보충된 둘베코스 변형된 이글즈 배지 (DMEM, 인비트로겐) 고 글루코스 중에서 성장시키고 유지시켰다. K562 세포는 10% 우태아 혈청 (FBS, 인비트로겐 15%) 및 페니실린/스트렙토마이신 (pen/strep, 인비트로겐)으로 보충된 RPMI (인비트로겐) 중에서 성장시키고 유지시켰다. 모든 세포를 습윤화된 인큐베이터 중 37℃ 및 5% CO2에서 유지시켰다.
HDR 효율을 검출하기 위해 안정한 293T 세포주를 참고 문헌 26 (상기 문헌은 그 전문이 본원에서 참조로 포함된다)에 기술되어 있는 바와 같이 확립시켰다. 구체적으로, 리포터 세포주는 종결 코돈의 삽입에 의해 파괴된 게놈적으로 통합된 GFP 코딩 서열 및 AAVS1 유전자좌로부터 유래된 68 bp 게놈 단편을 포함한다.
실시예 IV
리TALEN 활성 시험
293T 리포터 세포를 24 웰 플레이트 중 2 x 105개의 세포/웰의 밀도로 시딩하고, 리포텍트아민(Lipofectamine) 2000을 사용하여 제조사의 프로토콜에 따라 1 ㎍의 각각의 리TALEN 플라스미드 및 2 ㎍ DNA 도너 플라스미드로 형질감염시켰다. 형질감염 후 ~18 h 경과하였을 때, TrypLE 익스프레스 (인비트로겐)를 사용하여 세포를 수거하고, LSRFortessa 세포 분석기 (BD 바이오사이언시즈)를 사용하여 유세포 분석법 분석을 위해 200 ㎕ 배지 중에 재현탁시켰다. 유세포 분석법 데이터를 플로우조(FlowJo) (플로우조)를 이용하여 분석하였다. 각 형질감염 샘플당 25,000개 이상의 이벤트를 분석하였다. 293T에서의 내인성 AAVS1 유전자좌 표적화 실험을 위해, 형질감염 방법은 상기 기술된 바와 동일하였고, 형질감염 후 1주 경과하였을 때, 3 ㎍/ml의 약물 농도를 이용하여 퓨로마이신 선별을 수행하였다.
실시예 V
기능성 렌티바이러스 생성 평가
표준 PCR 및 클로닝 기법에 의해 렌티바이러스 벡터를 생성하였다. 렌티바이러스 플라스미드를 리포텍트아민 2000에 의해 렌티바이러스 패키징 믹스 (인비트로겐)를 이용하여 293FT 세포 (인비트로겐) 내로 형질감염시켜 렌티바이러스를 생성하였다. 형질감염 후 48 및 72 h 경과하였을 때, 상청액을 수집하고, 멸균 여과시키고, 100 ul 여과된 상청액을 폴리브렌과 함께 5 x 105개의 신선한 293T 세포에 첨가하였다. 하기 공식: 바이러스 적정량 = (GFP+ 293T 세포의 비율(%) * 형질도입하에 있는 초기 세포 개수)/(형질도입 실험에 사용된, 원래의 바이러스 수집 상청액 부피)에 기초하여 렌티바이러스 적정량을 계산하였다. 렌티바이러스의 기능성을 시험하기 위하여, 형질감염 후 3일 경과하였을 때, 렌티바이러스가 형질도입된 293T 세포를 리포텍트아민 2000 (인비트로겐)을 이용하여 mCherry 리포터를 포함하는 30 ng 플라스미드 및 500 ng pUC19 플라스미드로 형질감염시켰다. 형질감염 후 18시간 경과하였을 때, 악시오 옵저버(Axio Observer) Z.1 (자이스(Zeiss))을 이용하여 세포 영상을 분석하고, TrypLE 익스프레스 (인비트로겐)를 이용하여 수거하고, LSR포르테싸(LSRFortessa) 세포 분석기 (BD 바이오사이언시즈)를 이용하여 유세포 분석법 분석을 수행하기 위해 200 ㎕ 배지 중에 재현탁시켰다. BD FACS디바 (BD 바이오사이언시즈)를 이용하여 유세포 분석법 데이터를 분석하였다.
실시예 VI
리TALEN 및 Cas9-gRNA 게놈 편집 효율 시험
뉴클레오펙션 수행하기 2 h 전에 PGP1 iPSC를 Rho 키나제 (ROCK) 억제제 Y-27632 (Calbiochem) 중에서 배양하였다. P3 프라이머리 셀 4D-뉴클레오펙터 X 키트 (론자(Lonza))를 이용하여 형질감염을 수행하였다. 구체적으로, TrypLE 익스프레스 (인비트로겐)를 이용하여 세포를 수거하고, 16.4 ㎕ P3 뉴클레오펙터 용액, 3.6 ㎕ 보충물, 1 ㎍의 각각의 리TALEN 플라스미드 또는 1 ug Cas9 및 1 ug gRNA 구축물, 2 ㎕의 100 μM ssODN을 함유하는 20 ㎕ 뉴클레오펙션 혼합물 중에 2 x 106개의 세포를 재현탁시켰다. 이어서, 혼합물을 20 ㎕ 뉴클레오큐벳 스트립으로 옮기고, CB150 프로그램을 이용하여 뉴클레오펙션을 수행하였다. 세포를 처음 24 hr 동안 ROCK 억제제로 보충된 mTeSR1 배지 중 마트리겔 코팅된 플레이트 상에 플레이팅시켰다. dsDNA 도너를 이용하여 내인성 AAVS1 유전자좌 표적화 실험을 수행하는 경우, 동일한 방법을 수행하되, 단, 예외적으로, 2 ㎍ dsDNA 도너를 사용하였고, mTeSR1 배지를 형질감염 후 1주 경과하였을 때, 0.5 ug/mL 농도의 퓨로마이신으로 보충하였다.
본 실시예에서 사용된 리TALEN, gRNA 및 ssODN에 관한 정보는 하기 표 3 및 표 4에 열거되어 있다.
<표 3> 리TALEN 쌍/Cas9-gRNA 표적화 CCR5에 관한 정보
<표 4> ssODN-매개 게놈 편집 연구를 위한 ssODN 디자인
실시예 VII
표적화 영역의 앰플리콘 라이브러리 제조
뉴클레오펙션 후 6일 경과하였을 때, 세포를 수거하고, 0.1 ㎕ 프렙GEM(prepGEM) 조직 프로테아제 효소 (자이GEM(ZyGEM)) 및 1 ㎕ 프렙GEM 골드 완충제 (자이GEM)를 8.9 ㎕의 배지 중 2~5 X 105개의 세포에 첨가하였다. 이어서, 1 ul의 반응물을, 5 ul 2X 카파 히피 핫스타트 레디믹스(KAPA Hifi Hotstart Readymix) (카파 바이오시스템즈(KAPA Biosystems)) 및 100 nM 상응하는 증폭 프라이머 쌍을 함유하는 9 ㎕ PCR 믹스에 첨가하였다. 반응물을 95℃에서 5 min 동안, 이어서, 98℃, 20 s; 65℃, 20 s 및 72℃, 20 s의 15 사이클로 인큐베이션시켰다. 이어서, 사용되는 일루미나(Illumina) 서열 어댑터를 부가하기 위해, 5 ㎕ 반응 생성물을, 12.5 ㎕ 2X 카파 히피 핫스타트 레디믹스 (카파 바이오시스템즈) 및 200 nM의 일루미나 서열 어댑터를 포함하는 프라이머를 함유하는 20 ㎕의 PCR 믹스에 첨가하였다. 반응물을 95℃에서 5 min 동안, 이어서, 98℃, 20 s; 65℃, 20 s 및 72℃, 20 s의 25 사이클로 인큐베이션시켰다. QIA퀵 PCR 정제용 키트에 의해 PCR 생성물을 정제하고, 거의 동일한 농도로 혼합하고, MiSeq 퍼스널 시퀀서(MiSeq Personal Sequencer)를 이용하여 서열분석하였다. PCR 프라이머는 하기 표 5에 열거되어 있다.
<표 5> CCR5 표적화 부위 PCR 프라이머 서열
*인덱스-PCR 프라이머는 에피상트르 (스크립트Seq™ 인덱스 PCR 프라이머(ScriptSeq™ Index PCR Primers))로부터 구입
실시예 VIII
게놈 편집 평가 시스템 (GEAS)
희귀 게놈 변경을 검출하기 위해 차세대 서열분석을 사용하여 왔다. 참고 문헌 27-30 (상기 문헌들은 그 전문이 본원에서 참조로 포함된다)을 참조할 수 있다. 상기 접근법을 광범위하게 사용함으로써 hiPSCS 중 HDR 및 NHEJ 효율을 신속하게 평가할 수 있도록 하기 위해, "파이프라인"으로 지칭되는 소프트웨어를 작성하여 게놈 조작 데이터를 분석하였다. 상기 파이프라인은 상이한 도구, 예컨대, R, BLAT, 및 FASTX 툴키트(FASTX Toolkit)를 사용하는 하나의 단일 유닉스(Unix) 모듈로 통합된다.
바코드 분할: 샘플 군을 함께 풀링하고, MiSeq 150 bp 쌍 형성 단부 (PE150) (일루미나 넥스트 젠 시퀀싱(Illumina Next Gen Sequencing))를 이용하여 서열분석한 후, 이어서, FASTX 툴키트를 이용하여 DNA 바코드에 기초하여 분류하였다.
품질 여과: 서열 품질이 더 낮은 (프레드 점수<20) 뉴클레오티드를 트리밍하였다. 트리밍한 후, 길이가 80개의 뉴클레오티드보다 더 짧은 리드는 폐기하였다.
지도화: BLAT를 이용하여 쌍 형성 리드를 독립적으로 참조 게놈으로 지도화하고, 출력값으로서 .psl 파일을 작성하였다.
Indel 호출: Indel은 정렬 중 2 블록의 매치를 함유하는 전장의 리드로서 정의되었다. 2개의 쌍 형성 단부 리드 모두에서 하기 패턴 다음의 리드만이 고려되었다. 품질 관리자로서 indel 리드는 참조 게놈과 매칭되는 최소 70 nt의 블록을 가져야 하고, 두 블록은 모두 길이가 20 nt 이상이어야 한다. 참조 게놈에 대해 상대적인 각 블록의 위치에 의해 indel의 크기 및 위치를 계산하였다. indel을 함유하는 리드의 비율(%) (하기 방정식 1 참조)로서 비-상동성 단부 연결 (NHEJ)을 추정하였다. NHEJ 이벤트의 대다수는 표적화 부위 인접부에서 검출되었다.
상동성 지정 재조합 (HDR) 효율: DSB 상의 중심부의 12 bp 윈도우 내의 패턴 매칭 (grep)을 이용하여 참조 서열을 함유하는 리드, 참조 서열의 변형 (2 bp 의도된 미스매치)을 함유하는 리드, 및 2 bp 의도된 미스매치 내에 오직 1 bp 돌연변이만을 함유하는 리드에 상응하는 특이 시그니처를 계수하였다 (하기 방정식 1 참조).
방정식 1. NHEJ 및 HDR 추정
A= 참조와 동일한 리드: XXXXXABXXXXX
B= ssODN에 의해 프로그램화된 2 bp 미스매치를 함유하는 리드: XXXXXabXXXXX
C= 표적 부위 중 오직 1 bp 돌연변이만을 함유하는 리드: 예컨대, XXXXXaBXXXXX 또는 XXXXXAbXXXXX
D = 상기 기술된 바와 같은 indel을 함유하는 리드
실시예 IX
전이증식된 hiPSC의 유전자형 스크리닝
FACS 분류 전 적어도 2시간 동안 SMC4 (5 uM 티아조비빈, 1 uM CHIR99021, 0.4 uM PD0325901, 2 uM SB431542) 로 보충된 mTesr-1 배지로 피더 세포 무함유 배양물 상의 인간 iPS 세포를 미리 처리하였다 (참고 문헌 23 (상기 문헌은 그 전문이 본원에서 참조로 포함된다) 참조). 아큐타제(Accutase) (밀리포어(Millipore))를 이용하여 배양물을 해리시키고, SMC4 및 1~2 X 107 /mL 농도의 생존력 염료 ToPro-3 (인비트로겐)으로 보충된 mTesr-1 배지 중에 재현탁시켰다. 멸균 조건하에서 100 um 노즐이 장착된 BD FACSAria II SORP UV (BD 바이오사이언시즈)를 이용하여 생 hiPS 세포를 방사선 조사된 CF-1 마우스 배아 섬유아세포 (글로벌 스템(Global Stem))으로 코팅된 96 웰 플레이트로 단일 세포 분류하였다. 각 웰은 SMC4 및 5 ug/ml 피브로넥틴 (시그마(Sigma))으로 보충된, 100 ng/ml 재조합 인간 염기성 섬유아세포 성장 인자 (bFGF) (밀리포어)를 포함하는 hES 세포 배지를 함유하였다 (참고 문헌 31 (상기 문헌은 그 전문이 본원에서 참조로 포함된다) 참조). 분류 후, 플레이트를 3 min 동안 70 x g로 원심분리하였다. 분류 후 4일째 콜로니 형성을 관찰할 수 있었으며, 배양 배지를 SMC4가 보충된 hES 세포 배지로 교체하였다. 분류 후 8일 경과하였을 때, SMC4를 hES 세포 배지로부터 제거할 수 있다.
형광 활성화된 세포 분류 (FACS) 후 8일 경과하였을 때, 수천 개의 세포를 수거하고, 0.1 ul 프렙GEM 조직 프로테아제 효소 (자이GEM) 및 1 ul 프렙GEM 골드 완충제 (자이GEM)를 8.9 ㎕의 배지 중 세포에 첨가하였다. 이어서, 반응물을, 35.5 ml 백금 1.1X 슈퍼믹스(Supermix) (인비트로겐), 250 nM의 각 dNTP 및 400 nM 프라이머를 함유하는 40 ㎕ PCR 믹스에 첨가하였다. 반응물을 95℃에서 3 min 동안, 이어서, 95℃, 20 s; 65℃, 30 s 및 72℃, 20 s의 30 사이클로 인큐베이션시켰다. 생성물을 표 5의 PCR 프라이머 중 하나를 이용하여 생어 서열분석하고, DNASTAR (DNASTAR)을 이용하여 서열을 분석하였다.
실시예 X
hiPSC의 면역염색 및 테라토마 검정
세포를 하기 항체: 항-SSEA-4 PE (밀리포어) (1:500으로 희석); Tra-1-60 (BD 파미겐(BD Pharmingen)) (1:100으로 희석)를 이용하여 37℃에서 60분 동안 넉아웃 DMEM/F-12 배지 중에서 인큐베이션시켰다. 인큐베이션 후, 세포를 넉아웃 DMEM/F-12로 3회에 걸쳐 세척하고, 악시오 옵저버 Z.1 (자이스) 상에서 영상화하였다.
테라토마 형성 분석을 수행하기 위해, 콜라게나제 IV형 (인비트로겐)을 이용하여 인간 iPSC를 수거하고, 세포를 200 ㎕ 마트리겔에 재현탁시키고, Rag2감마 녹아웃 마우스의 뒷다리 내로 근육내로 주사하였다. 주사 후 4-8주 경과하였을 때, 테라토마를 단리시키고, 포르말린 중에 고정시켰다. 이어서, 테라토마를 헤마톡실린 및 에오신 염색에 의해 분석하였다.
실시예 XI
리TALEN을 이용한 인간 체세포 및 인간 줄기 세포 중 게놈 유전자좌 표적화
특정 측면에 따라, 통상의 기술자에게 공지된 TALE를 변형 또는 재코딩하여 반복부 서열을 제거한다. 본원에 기술된 바이러스 전달 비히클에서 및 각종 세포주 및 유기체에서의 게놈 편집 방법에서 변형 및 사용에 적합한 상기 TALE는 참고 문헌 2, 7-12 (상기 문헌들은 그 전문이 본원에서 참조로 포함된다)에 개시되어 있다. 반복적 TALE RVD 어레이 서열을 조립하는 수개의 전략법이 개발되었다 (참고 문헌 14 및 32-34 (상기 문헌들은 그 전문이 본원에서 참조로 포함된다)를 참조). 그러나, 일단 조립되고 나면, TALE 서열 반복부는 불안정한 상태 그대로 유지되며, 이로 인해 상기 도구의 광범위한 유용성은 제한되고, 특히 바이러스 유전자 전달 비히클인 경우에 그러하다 (참고 문헌 13 및 35 (상기 문헌들은 그 전문이 본원에서 참조로 포함된다)를 참조). 따라서, 본 개시내용의 한 측면은 반복부가 결핍된, 예컨대, 반복부가 완전히 TALEN에 관한 것이다. 상기 재코딩된 TALE는 그를 통해서 확장된 TALE RVD 어레이를 더욱 신속하고, 간단하게 합성할 수 있기 때문에 이롭다.
반복부를 제거하기 위하여, TALE RVD 어레이의 뉴클레오티드 서열을 연산적으로 진화시켜 아미노산 조성은 유지시키면서, 서열 반복부의 개수를 최소화시켰다. 16개의 일렬 RVD DNA 인식 단량체와, 마지막 절반 RVD 반복부를 코딩하는 재코딩된 TALE (리TALE)에는 임의의 12 bp 반복부가 없다 (도 5a 참조). 특히, 이러한 재코딩 수준은 전장의 리TALE 구축물로부터 임의의 특이 단량체 또는 서브섹션을 PCR 증폭시키는 데 충분하다 (도 5b 참조). 반복부 중쇄 서열과 관련된 추가 비용 또는 방법을 초래하지 않으면서, 표준 DNA 합성 기술을 사용하여 개선된 디자인의 리TALE를 합성할 수 있다 (참고 문헌 36 (상기 문헌은 그 전문이 본원에서 참조로 포함된다) 참조). 추가로, 재코딩된 서열 디자인을 통해서는 본원에 기술된 방법에 기술된 바와 같이, 및 도 6을 참조하여 변형된 등온 조립 반응을 이용함으로써 리TALE 구축물을 효율적으로 조립할 수 있다.
게놈 편집 NGS 데이터를 하기와 같이 통계학적으로 분석하였다. HDR 특이성 분석을 위해, 정확한 이항 시험을 사용하여 2 bp 미스매치를 함유하는 다양한 개수의 서열 리드를 관찰할 수 있는 확률을 계산하였다. 부위 표적화 전후의 10 bp 윈도우에 관한 서열분석 결과에 기초하여, 두 윈도우의 최대 염기 변화율 (P1 및 P2)을 추정하였다. 두 표적 bp 각각의 변화는 독립적이라는 널 가설을 사용하여, 우연히 표적화 부위에서 2 bp 미스매치를 관찰할 수 있는 예상 확률은 상기 두 확률의 곱 (P1*P2)으로서 계산하였다. 총 리드 개수 N 및 HDR 리드 개수 n을 함유하는 데이터세트 전제하에, 본 발명자들은 관찰된 HDR 효율의 p 값을 계산하였다. HDR 감도 분석을 위해, ssODN DNA 도너는 표적화 게놈에 대하여 2 bp 미스매치를 함유하였으며, 그 결과, ssODN이 표적화 게놈 내로 도입될 경우, 가능하게는 2개의 표적 bp 중 염기 변이가 공동으로 존재하게 되었다. 다른 의도되지 않은 관찰된 서열 변이는 가능하게는 동시에 바뀌지 않을 것이다. 따라서, 의도되지 않은 변이는 상호의존성이 훨씬 더 적었다. 이러한 가정하에, 상호 정보 (MI)를 이용하여 모든 다른 위치 쌍에서의 동시의 두 염기쌍 변이의 상호 의존성을 측정하고, 2 bp 부위를 표적화하는 것의 MI가 모든 다른 위치 쌍의 MI보다 높은 최소 HDR로서 HDR 검출 한계를 추정하였다. 주어진 실험의 경우, 원 fastq 파일로부터의 의도된 2 bp 미스매치를 포함하는 HDR 리드를 확인하였고, 원래의 데이터 세트로부터 상이한 개수의 HDR 리드를 체계적으로 제거함으로써 HDR 효율이 희석된 fastq 파일 세트를 모의하였다. 표적화 부위를 중심으로 한 20 bp의 윈도우 내의 모든 위치 쌍 사이의 상호 정보 (MI)를 계산하였다. 상기 계산에서는 임의의 두 위치 사이의 염기 조성에 관한 상호 정보를 계산한다. 상기 기술된 HDR 특이성 척도와 달리, 상기 척도는 임의의 특정 표적 염기 쌍으로 변이될 수 있는 위치 쌍의 경향을 평가하는 것이 아니라, 오직 동시에 변이될 수 있는 그의 경향만을 평가할 수 있다 (도 8a). 하기 표 6은 CCR5를 표적화하는 리TALEN/ssODN의 HDR 및 NHEJ 효율 및 Cas9-gRNA의 NHEL 효율을 보여주는 것이다. 본 발명자들은 패키지 이포테오(infotheo)를 이용하여 R 및 MI에 관한 본 발명자들의 분석을 계산하였다.
<표 6>
* HDR 검출 한계가 검출된 실제 HDR을 초과하는 군
편집 효율과 후성적 상태 사이의 상관관계는 하기와 같이 처리하였다. 피어슨 상관관계 계수를 계산하여 후성적 파라미터 (DNase I HS 또는 뉴클레오좀 점유) 및 게놈 조작 효율 (HDR, NHEJ) 사이의 가능한 연관성을 연구하였다. UCSC 게놈 브라우저 (hiPSC DNase I HS: /gbdb/hg19/bbi/wgEncodeOpenChromDnaseIpsnihi7Sig.bigWig)로부터 DNAaseI 하이퍼센시티비티(DNAaseI Hypersensitivity) 데이터세트를 다운로드 받았다.
P 값을 계산하기 위해, 후성적 파라미터의 위치를 임의 추출하여 (N=100,000) 구축된 모의 분포와 관찰된 상관관계를 비교하였다. 관찰된 상관관계가 모의 분포의 95번째 백분위수보다 더 높거나, 또는 5번째 백분위수보다 더 낮은 것은 잠재적으로 연관성이 있는 것으로 간주되었다.
인간 세포에서 상응하는 재코딩되지 않는 TALEN과 비교하여 리TALEN의 기능을 측정하였다. 참고 문헌 37 (상기 문헌은 그 전문이 본원에서 참조로 포함된다)에 기술된 바와 같이 프레임 쉬프팅 삽입을 보유하는 GFP 리포터 카세트를 함유하는 HEK 293 세포주를 사용하였다. 도 1a 또한 참조할 수 있다. 프로모터가 없는 GFP 도너 구축물과 함께 삽입 서열을 표적화하는 TALEN 또는 리TALEN을 전달하면, GFP 카세트의 DSB 유도성 HDR 수복이 이루어지고, 이로써, GFP 수복 효율은 뉴클레아제 절단 효율을 평가하는 데 사용될 수 있다. 참고 문헌 38 (상기 문헌은 그 전문이 본원에서 참조로 포함된다)을 참조할 수 있다. 리TALEN은 TALEN에 의해 달성된 것과 유사한 정도로 (1.2%), 형질감염된 세포의 1.4%로 GFP 수복을 유도하였다 (도 1b 참조). PGP1 hiPSC 중 AAVS1 유전자좌에서의 리TALEN의 활성을 시험하고 (도 1c 참조), 특이 삽입을 함유하는 세포 클론을 성공적으로 회수함으로써 (도 1d, e 참조), 리TALEN이 체세포 및 다능성 인간 세포, 둘 모두에서 활성임을 확인할 수 있었다.
반복부를 제거함으로써 리TALE 카고를 포함하는 기능성 렌티바이러스를 생성할 수 있었다. 구체적으로, 리TALE-2A-GFP를 코딩하는 렌티바이러스 입자를 패키징하고, 렌티-리TALE-2A-GFP 감염된 293T 세포 풀로 mCherry 리포터를 형질감염시켜 바이러스 입즈에 의해 코딩된 리TALE-TF의 활성을 시험하였다. 렌티-리TALE-TF가 형질도입된 293T 세포는 오직 리포터만이 있는 음성의 것과 비교하였을 때 36X 리포터 발현 활성화를 보였다 (도 7a,b,c 참조). 렌티바이러스 감염된 세포에서의 리TALE-TF의 서열 완전성을 체크하였고, 시험된 10개의 클론 모두에서 전장의 리TALE가 검출되었다 (도 7d 참조).
실시예 XII
게놈 편집 평가 시스템 (GEAS)을 이용한 hiPSC에서의 리TALE 및 Cas9-gRNA 효율 비교
hiPSC에서의 리TALEN 대 Cas9-gRNA의 편집 효율을 비교하기 위하여, NHEJ 및 HDR 유전자 편집 이벤트, 둘 모두를 확인하고, 정량화하는 차세대 서열분석 플랫폼 (게놈 편집 평가 시스템)을 개발하였다. 2 bp 미스매치를 제외하면 표적 부위와 동일한 90nt ssODN 도너와 함께, CCR5의 상류 영역 (표 3에서 리TALEN, Cas9-gRNA 쌍 #3)을 표적화하는 리TALEN 쌍 및 Cas9-gRNA를 디자인하고, 구축하였다 (도 2a 참조). 뉴클레아제 구축물 및 도너 ssODN을 hiPSC 내로 형질감염시켰다. 유전자 편집 효율을 정량화하기 위해, 형질감염 후 3일 경과하였을 때, 표적 게놈 영역에 대한 쌍 형성 단부에 관한 심층 서열분석을 수행하였다. 정확한 2 bp 미스매치를 함유하는 리드의 비율(%)에 의해 HDR 효율을 측정하였다. NHEJ 효율은 indel을 보유하는 리드의 비율(%)에 의해 측정하였다.
hiPSC 내로 ssODN만을 전달한 결과, 최소 비율의 HDR 및 NHEJ를 얻은 반면, 리TALEN 및 ssODN을 전달하였을 때 HDR 효율은 1.7%이고, NHEJ 효율은 1.2%였다 (도 2b 참조). ssODN과 함께 Cas9-gRNA를 도입하였을 때 HDR 효율은 1.2%이고, NHEJ 효율은 3.4%였다. 특히, 게놈 결실 및 삽입의 비율은 두 리TALEN 결합 부위 사이의 스페이서 영역 중간에서 가장 컸지만, Cas9-gRNA 표적화 부위의 프로토스페이서 결합 모티프 (PAM) 서열 상류의 3-4 bp에서 가장 컸는데 (도 2b 참조), 이는 이중 가닥 절단이 상기 영역에서 발생하는 바, 그에 따라 예상되는 바와 같은 것이었다. 리TALEN에 의해 생성된 게놈 결실 크기의 중간값은 6 bp이고, 삽입 크기의 중간값은 3 bp인 것으로 관찰되었고, Cas9-gRNA에 의한 게놈 결실 크기의 중간값은 7 bp이고, 삽입 크기의 중간값은 1 bp인 것으로 관찰되었으며 (도 2b 참조), 이는 일반적으로 NHEJ에 의해 생성된 DNA 손상 패턴과 일치하는 것이었다 (참고 문헌 4 (상기 문헌은 그 전문이 본원에서 참조로 포함된다) 참조). 차세대 서열분석 플랫폼의 수회의 분석을 통해 GEAS는 0.007%만큼 낮은 HDR 검출율을 검출할 수 있는 것로 나타났는데, 이는 고도로 재현가능할 뿐만 아니라 (복제물 사이의 변동 계수 = ± 15% * 측정된 효율), 가장 일반적으로 사용되는 미스매치 감도성 엔도뉴클레아제 검정법보다 400X 더 큰 고감도성이다 (도 8 참조).
편집 효율을 측정하기 위하여 CCR5 게놈 유전자좌에서 15개의 부위로 표적화되는 리TALEN 쌍 및 Cas9-gRNA를 구축하였다 (도 2c 참조, 표 3 참조). 광범위한 DNaseI 감도를 나타내기 위하여 상기 부위를 선택하였다 (참고 문헌 39 (상기 문헌은 그 전문이 본원에서 참조로 포함된다) 참조). 상응하는 ssODN 도너를 이용하여 (표 3 참조) 뉴클레아제 구축물을 PGP1 hiPSC 내로 형질감염시켰다. 형질감염 후 6일 경과하였을 때, 상기 부위에서의 게놈 편집 효율을 프로파일링하였다 (하기 표 6). ssODN 도너와 함께 15개의 리TALEN 쌍 중 13개의 경우, NHEJ 및 HDR은 통계학적 검출 역치보다 큰 수준으로 검출되었고, 평균 NHEJ 효율은 0.4%이고, 평균 HDR 효율은 0.6%였다 (도 2c 참조). 추가로, 같은 표적화 유전자좌에서의 HR과 NHEJ 효율 사이에 통계학상 유의적인 양의 상관관계 (r2 =0.81)가 있는 것으로 밝혀졌고 (P<1 X 10-4) (도 9a 참조), 이는 HDR과 NHEJ, 둘 모두의 공통된 상류 단계인 DSB 생성이 리TALEN-매개 게놈 편집을 위한 속도 제한 단계임을 제안하는 것이다.
대조적으로, 15개의 Cas9-gRNA 쌍은 모두 평균 NHEJ 효율 3% 및 평균 HDR 효율 1.0%로, 유의적인 수준의 NHEJ 및 HR을 보였다 (도 2c 참조). 추가로, Cas9-gRNA에 의해 도입된 NHEJ 및 HDR 효율 사이에 양의 상관관계 또한 검출되었으며 (도 9b 참조) (r2=0.52, p=0.003), 이는 리TALEN에 대한 관찰결과와 일치하는 것이었다. Cas9-gRNA에 의해 달성된 NHEJ 효율은 리TALEN에 의해 달성된 것보다 유의적으로 더 높았다 (t 검정, 쌍 형성 단부, P=0.02). gRNA 표적화 서열의 용융 온도와 NHEJ 효율 사이의, 중간 정도이지만, 통계학상 유의적인 상관관계가 관찰되었는데 (도 9c 참조) (r2=0.28, p=0.04), 이는 gRNA 및 그의 게놈 표적 사이의 염기쌍 형성의 강도가 Cas9-gRNA-매개 DSB 생성 효율에서 편차가 28% 정도라는 것을 설명할 수 있다는 것을 제안한다. 비록 Cas9-gRNA가 상응하는 리TALEN보다 평균 7배 더 높은 NHEJ 수준을 생성하기는 하지만, Cas9-gRNA는 단지 상응하는 리TALEN의 것 (평균 = 0.6%)과 유사한 HDR 수준을 달성하였다 (평균=1.0%). 과학적 이론으로 한정하고자 하지 않으면서, 본 결과는 DSB에서의 ssODN 농도가 HDR에 대한 제한 인자이거나, 또는 Cas9-gRNA에 의해 생성된 게놈 절단 구조가 효과적인 HDR에 대하여 바람직하지 않다는 것을 제안하는 것일 수 있다. 어느 방법의 경우에도 DNaseI HS와 게놈 표적화 효율 사이에는 어떤 상관관계도 관찰되지 않았다 (도 10 참조).
실시예 XIII
HDR을 위한 ssODN 도너 디자인의 최적화
hiPSC에서의 고성능 ssODN을 하기와 같이 디자인하였다. 모두가 CCR5 리TALEN 쌍 #3 표적 부위의 스페이서 영역의 한가운데 동일한 2 bp 미스매치를 보유하는 것인, 상이한 길이의 (50-170 nt) ssODN 도너 세트를 디자인하였다. HDR 효율은 ssODN 길이에 따라 달라지는 것으로 관찰되었고, 90 nt ssODN의 경우, 최적의 HDR 효율은 ~1.8%인 것을 관찰된 반면, 그보다 더 긴 ssODN은 HDR 효율을 감소시켰다 (도 3a 참조). dsDNA 도너가 뉴클레아제와 함께 사용될 때, 더욱 긴 상동성 영역은 HDR 비율을 개선시키기 때문에 (참고 문헌 40 (상기 문헌은 그 전문이 본원에서 참조로 포함된다) 참고), 본 결과에 대하여 가능한 이유는 ssODN이 선택적 게놈 수복 프로세스에서 사용될 수 있고; 더욱 긴 ssODN이 게놈 수복 장치에 덜 이용가능하거나; 또는 더욱 긴 ssODN은 dsDNA 도너에 비하여, 더욱 긴 상동성에 의해 획득되는 임의의 개선을 상쇄시키는 부정적 효과를 초래할 수 있다는 점이다 (참고 문헌 41 (상기 문헌은 그 전문이 본원에서 참조로 포함된다) 참조). 그러나, 상기의 처음 두 이유 중 어느 것이 되는 경우, 이때 NHEJ 비율은, NHEJ 수복이 ssODN 도너를 포함하지 않기 때문에, 영향을 받지 않거나, 또는 ssODN이 더 길어짐에 따라 증가하여야 한다. 그러나, NHEJ 비율은 HDR과 함꼐 감소하는 것으로 관찰되었으며 (도 3a 참조), 이는 더욱 긴 ssODN이 상쇄 효과를 나타낸다는 것을 제안한다. 더욱 긴 ssODN이 세포에 독성이거나 (참고 문헌 42 (상기 문헌은 그 전문이 본원에서 참조로 포함된다) 참조)), 또는 더욱 긴 ssODN의 형질감염이 DNA 프로세싱 기구를 포화시킴으로써 DNA 몰 흡수량을 감소시키고, 리TALEN 플라스미드를 흡수하거나, 그를 발현시키는 세포의 능력을 감소시킨다는 것이 가능한 가설이 될 것이다.
ssODN 도너가 보유하는 미스매치의 도입 비율이 그의 이중 가닥 절단 ("DSB")까지의 거리에 따라 어떻게 달라지는지를 조사하였다. 모두가 리TALEN 쌍 #3의 스페이서 영역의 중앙에 동일한 2 bp 미스매치 (A)를 포함하는 일련의 90 nt ssODN을 디자인하였다. 각 ssODN은 또한 중앙부로부터 다른 거리에 제2의 2 bp 미스매치 (B)를 함유하였다 (도 3b 참조). 오직 중앙부의 2 bp 미스매치만을 포함하는 ssODN을 대조군으로 사용하였다. 상기 ssODN을 각각 리TALEN 쌍 #3과 함께 개별적으로 도입하였고, 그 결과는 GEAS를 이용하여 분석하였다. A 미스매치가 도입된 (A만 또는 A+B) 비율에 의해 측정된 바, 전체 HDR은 B 미스매치가 중앙부로부터 더 멀어짐에 따라 감소하였다는 것을 (도 3b 참조, 도 11a 참조) 본 발명자들은 알 수 있었다. B가 A로부터 단지 10 bp만큼 떨어져 있을 때 관찰된 바 전체 HDR 비율이 더 높다는 것은 dsDNA 절단 바로 옆에 인접해 있는 게놈 DNA에 대해 ssODN이 어닐링될 필요가 더 적다는 것을 반영할 수 있다.
A로부터의 B까지의 각 거리의 경우, HDR 이벤트 중 일부는 오직 A 미스매치만을 도입한 반면, 또 다른 일부는 A 및 B 미스매치, 둘 모두를 도입하였다 (도 3b 참조 (A만 및 A+B)). 상기의 두 결과는 ssDNA 올리고 길이에 따른 유전자 전환 트랙에 기인하는 것일 수 있으며 (참고 문헌 43 (상기 문헌은 그 전문이 본원에서 참조로 포함된다) 참조), 이로서, A+B 미스매치의 도입은 B 미스매치 너머까지 확장된 긴 전환 트랙으로부터 초래되었고, 오직 A 미스매치만의 도입은 B에 도달하지 못한 더 짧은 트랙으로부터 초래되었다. 이러한 해석시, ssODN을 따른 양 방향의 유전자 전환 길이의 분포를 추정하였다 (도 11b 참조). 추정된 분포는 유전자 전환 트랙이 그의 길이가 증가함에 따라 점진적으로 덜 빈번하게 발생하게 된다는 것을 암시하는 것이며, 이러한 결과는 dsDNA 도너를 이용하였을 때 관찰된 유전자 전환 트랙 분포와 매우 유사한 결과로서, 단, dsDNA 도너의 경우에는 수백 개의 염기인데 반해, ssDNA 도너의 경우에는 수십개 염기 만큼의 고도로 압축된 거리 규모일 때이다. 상기 결과와 일치하는 것으로서, 중앙의 2 bp 미스매치 "A" 양측에 10 nt 만큼의 간격을 두고 이격되어 있는 3쌍의 2 bp 미스매치를 포함하는 ssODN을 사용하여 수행한 실험에서는 A만이 단독으로 도입된 경우의 패턴을 다른 경우에는 다중의 B 미스매치가 도입된 경우에는 그때의 것의 86%가 되도록 하였다 (도 11c). 비록 B만의 도입 이벤트 횟수가 너무 낮아 길이가 10 np 미만인 트랙 분포를 추정하기 어렵기는 했지만, 뉴클레아제 부위의 10 bp 이내의 짧은 트랙 영역이 지배적이라는 것은 명백하다 (도 11b 참조). 최종적으로, 단일 B 미스매치를 이용하여 수행한 모든 실험에서, 작은 분율의 B만의 도입 이벤트가 관찰되었으며 (0.04%~0.12%), 이는 대략적으로는 A로부터 B까지의 모든 길이 간에 걸쳐 일정하였다.
추가로, 여전히 도입은 관찰되면서, ssODN 도너가 리TALEN 유도 dsDNA 절단으로부터 얼마나 멀리 떨어져 배치될 수 있는지에 관하여 분석하였다. 리TALEN 유도 dsDNA 절단 부위로부터 더 큰 거리 범위 (-600 bp 내지 +400 bp) 만큼 떨어져 있는 부위를 표적화하는, 중앙 2 bp 미스매치를 포함하는 90 nt ssODN 세트를 시험하였다. ssODN이 ≥40 bp 만큼 떨어져 매칭될 때, 본 발명자들은 절단 영역에 걸쳐 중앙부에 위치한 대조군 ssODN과 비교하였을 때 >30x 더 낮은 HDR 효율을 관찰하였다 (도 3c 참조). 게놈이 ssDNA 도너 단독에 의해 변경된 실험에서 관찰된 바와 같이, 관찰된 낮은 수준의 도입은 dsDNA 절단과 관련이 없는 프로세스에 기인하는 것일 수 있다 (참고 문헌 42 (상기 문헌은 그 전문이 본원에서 참조로 포함된다) 참조). 한편, ssODN이 ~40 bp 만큼 떨어져 있을 때, 낮은 수준의 HDR이 존재하는 것은 dsDNA 절단의 미스매치를 함유하는 쪽에 위치하는 약화된 상동성과, dsDNA 절단의 나머지 다른 한쪽 상의 불충분한 ssODN 올리고 길이와의 조합에 기인하는 것일 수 있다.
Cas9-gRNA 매개 표적화를 위한 ssODN DNA 도너 디자인을 시험하였다. AAVS1 유전자좌를 표적화하는 Cas9-gRNA (C2)를 구축하고, 다양한 배향 (Oc: gRNA에 상보적 및 On: gRNA에 비상보적) 및 길이 (30, 50, 70, 90, 110 nt)의 ssODN 도너를 디자인하였다. Oc는 On보다 더 우수한 효율을 달성하였고, 70 mer Oc는 1.5%의 최적의 HDR 비율을 달성하였다 (도 3d 참조). ssODN과 함께 Cas9-유래 닉카제 (Cc: Cas9_D10A)에 의해 매개된 HDR 효율이 C2보다 유의적으로 더 낮았다는 사실에도 불구하고 (t 검정, 쌍 형성 단부, P=0.02), Cc를 사용하였을 때, 같은 ssODN 가닥 편향성이 검출되었다 (도 12 참조).
실시예 XIV
교정된 세포의 hiPSC 클론 단리
GEAS를 통해 리TALEN 쌍 #3이 hiPSC에서, 정확하게 편집된 세포가 일반적으로 클론 스크리닝에 의해 단리될 수 있는 수준인 ~1%의 효율로 정확한 게놈 편집을 달성하였다는 것이 밝혀졌다. HiPSC는 단일 세포만큼 열등한 생존 능력을 가진다. 단일 세포 FACS 분류 방법과 함께 참고 문헌 23 (상기 문헌은 그 전문이 본원에서 참조로 포함된다)에 기술된 최적화된 프로토콜을 사용하여 단일 hiPSC 분류 및 유지를 위한 강건한 플랫폼을 확립하였고, 여기서, hiPSC 클론은 >25%의 생존율로 회수될 수 있다. 상기 방법을 신속하고 효율적인 유전형 분석 시스템과 조합하여 1시간 이내에 단일 튜브 반응에서 염색체 DNA 추출 및 표적화된 게놈 증폭을 수행하였으며, 이로써 편집된 hiPSC의 대규모 유전형 분석을 수행할 수 있었다. 동시에, 상기 방법은 선별 없이 게놈 편집된 hiPSC를 강건하게 수득하기 위한 파이프라인을 포함한다.
본 시스템을 입증하기 위하여 (도 4a 참조), PGP1 hiPSC를 부위 #3에서 ssODN 표적화 CCR5 및 리TALEN 쌍으로 형질감염시켰다 (표 3 참조). 형질감염된 세포 중 일부를 이용하여 GEAS를 수행하였고, 이 결과, HDR 빈도는 1.7%인 것으로 나타났다 (도 4b 참조). 분류된 단일 세포 클론의 회수율이 25%라는 것과 함께, 상기의 정보를 통해 5개의 96 웰 플레이트로부터 1개 이상의 정확하게 편집된 클론을 98%의 포아송 확률(Poisson probability) (μ=0.017*0.25*96*5*2로 가정할 때)로 수득할 수 있다고 추정할 수 있다. 형질감염 후 6일 경과하였을 때, hiPSC를 FACS 분류하고, 분류 후 8일 경과하였을 때, 100개의 hiPSC 클론을 스크리닝하였다. 생어 서열분석 결과, 상기의 비선별된 hiPSC 콜로니 100개 중 2개가 ssODN 도너에 의해 도입된 2 bp 돌연변이를 포함하는 이형접하성 유전자형을 함유하는 것으로 밝혀졌다 (도 4c 참조). 1% (1%=2/2*100, 스크리닝된 100개의 세포 중 2개의 단일 대립유전자 교정 클론)의 표적화 효율은 차세대 서열분석 분석 (1.7%)과 일치하는 것이었다 (도 4b 참조). 생성된 hiPSC의 다능성은 SSEA4 및 TRA-1-60에 대한 면역염색법으로 확인하였다 (도 4d 참조). 성공적으로 표적화된 hiPSC 클론은 3개의 배엽 모두를 특징으로 하는 성숙한 테라토마를 생성할 수 있었다 (도 4e 참조).
실시예 XV
연속적인 세포 게놈 편집 방법
특정 측면에 따라, 세포가 표적 DNA에 상보적인 RNA와 공동 국재화 복합체를 형성하고, 부위 특이적인 방식으로 표적 DNA를 절단하는 효소를 코딩하는 핵산을 포함하도록 유전적으로 변형된 것인, 인간 세포, 예를 들어, 인간 줄기 세포를 비롯한 세포에서의 게놈 편집 방법을 제공한다. 상기 효소는 RNA에 의해 가이드되는 DNA 결합 단백질, 예컨대, II형 CRISPR 시스템의 RNA에 의해 가이드되는 DNA 결합 단백질을 포함한다. 예시적인 효소는 Cas9이다. 본 측면에 따라, 세포는 효소를 발현하고, 세포 주변 배지로부터 세포에 가이드 RNA를 제공한다. 가이드 RNA 및 효소는 효소가 DNA를 절단하는 곳인 표적 DNA에서 공동 국재화 복합체를 형성한다. 임의적으로, 도너 핵산은 예를 들어, 비상동성 단부 연결 또는 상동성 재조합에 의해 절단 부위에서 DNA 내로의 삽입을 위해 존재할 수 있다. 한 측면에 따라, 표적 DNA에 상보적인 RNA와 공동 국재화 복합체를 형성하고, 부위 특이적인 방식으로 표적 DNA를 절단하는 효소, 예컨대, Cas9를 코딩하는 핵산은 프로모터의 영향하에 있으며, 예컨대, 핵산은 활성화되고, 침묵화될 수 있다. 상기 프로모터는 통상의 기술자에게 주지되어 있다. 예시적인 한 프로모터는 dox 유도성 프로모터이다. 한 측면에 따라, 세포는 표적 DNA에 상보적인 RNA와 공동 국재화 복합체를 형성하고, 부위 특이적인 방식으로 표적 DNA를 절단하는 효소를 코딩하는 핵산을 그의 게놈 내로 가역적으로 삽입함으로써 유전적으로 변형된다. 일단 삽입되고 나면, 핵산은 시약, 예컨대, 트랜스포사제의 사용에 의해 제거될 수 있다. 상기 방식으로, 핵산은 사용 이후에 쉽게 제거될 수 있다.
한 측면에 따라, CRISPR 시스템을 이용한 인간 유도 다능성 줄기 세포 (hiPSC)에서의 연속적인 게놈 편집 시스템을 제공한다. 예시적인 측면에 따라, 본 방법은 Cas9가 게놈 내에 가역적으로 삽입되어 있는 hiPSC 세포주 (Cas9-hiPSC); 및 그의 천연 형태로부터 Cas9와 함께 사용될 수 있도록 세포 주변 배지로부터 세포로 그가 통과할 수 있도록 변형된 gRNA를 이용하는 것을 포함한다. 포스페이트 기를 제거하는 방식으로 포스파타제를 이용하여 상기 gRNA를 처리하였다. 세포에서의 게놈 편집은 Cas9와 함께 조직 배양 배지 중 포스파타제로 처리된 gRNA를 보충함으로써 수행된다. 본 접근법을 통해서 현재까지 보고된 최고 효율보다 2-10X 배 더 효율적인 방식으로, 단일 처리일인 경우에는 최대 50%까지의 효율로 HiPSC에서 흔적 없는 게놈 편집을 수행할 수 있다. 추가로, 본 방법은 사용이 용이하고, 세포 독성이 유의적으로 더 낮다. 본 개시내용의 실시양태는 생물학적 연구 및 진단 적용을 위한 hiPSC의 단일 편집, 생물학적 연구 및 진단 적용을 위한 hiPSC의 다중 편집, hiPSC의 지향적 진화 및 hiPSC 및 그의 유도체 세포의 표현형 스크리닝을 포함한다.
특정 측면에 따라, 줄기 세포 이외에도 본원에 기술된 다른 세포주 및 유기체가 사용될 수 있다. 예를 들어, 안정한 Cas9 통합 마우스 세포 및 래트 세포를 생성할 수 있고, 포스파타제 처리된 gRNA를 세포 주변 배지로부터 국소적으로 도입함으로써 조직 특이 게놈 편집을 수행할 수 있도록 동물 세포, 예컨대, 마우스 또는 래트 세포에 대해 본원에 기술된 방법이 사용될 수 있다. 또한, 다른 Cas9 유도체가 다수의 세포주 및 유기체 내로 삽입될 수 있고, 표적화된 게놈 조작, 예컨대, 서열 특이 니킹, 유전자 활성화, 억제 및 후성적 변형이 수행될 수 있다.
본 개시내용의 측면은 Cas9가 게놈 내로 삽입된 안정한 hiPSC를 제조하는 것에 관한 것이다. 본 개시내용의 측면은 세포벽을 통해 세포 내로 진입할 수 있고, 세포의 면역 반응을 피하면서 Cas9와 공동 국재화할 수 있도록 RNA를 변형시키는 것에 관한 것이다. 상기 변형된 가이드 RNA를 최소의 독성으로 최적의 형질감염 효율을 달성할 수 있다. 본 개시내용의 측면은 포스파타제 처리된 gRNA를 사용한 Cas9-hiPSC에서의 최적화된 게놈 편집에 관한 것이다. 본 개시내용의 측면은 hiPSC로부터 Cas9를 제거하여 흔적 없는 게놈 편집을 달성하고, 여기서, Cas9를 코딩하는 핵산은 게놈 내로 가역적으로 배치된 것인 것을 포함한다. 본 개시내용의 측면은 원하는 유전자 돌연변이를 생성하기 위해 Cas9가 게놈 내로 삽입된 hiPSC를 사용하는 생체 의학적 조작을 포함한다. 상기 조작된 hiPSC는 다능성을 유지하고, 환자 세포주의 표현형을 완전하게 재현하는, 심장 근육 세포를 비롯한 각종 세포 유형으로 성공적으로 분화될 수 있다.
본 개시내용의 측면은 다중 게놈 편집을 위한 포스파타제 처리된 gRNA 라입러리를 포함한다. 본 개시내용의 측면은 약물 스크리닝을 위한 자원으로서의 역할을 할 수 있는 것인, 각각의 것이 1개를 게놈 중 수개의 지정된 돌연변이를 운반하는 PGP 세포주의 라이브러리를 생성하는 것을 포함한다. 본 개시내용의 측면은 레트로트랜스요소 모두가 상기 요소의 위치 및 활성을 추적하기 위해 상이한 서열로 바코딩된 것인 PGP1 세포주를 생성하는 것을 포함한다.
실시예 XVI
Cas9가 게놈 내로 삽입된 안정한 hiPSC 생성
dox 유도성 프로모터 하에 Cas9를 코딩하고, 피기백 트랜스포사제의 도움으로 게놈 내로 삽입되고, 그에서 제거될 수 있는 피기백 벡터 내로 구축물을 배치하였다. PCR 반응을 통해 벡터의 안정한 삽입을 확인하였다 (도 14 참조). RT-QPCR을 통해 유도성 Cas9 발현을 측정하였다. 배양 배지 중 1 ug/mL DOX 보충 후 8시간 경과하였을 때, Cas9의 mRNA 수준은 1,000X 증가하였고, DOX 제거 후 ~20시간 경과시 Cas9 mRNA 수준은 정상 수준으로 하락하였다 (도 15 참조).
한 측면에 따라, Cas9-hiPSC 시스템 기반 게놈 편집은 hiPSC에서의 형질감염 효율이 보통 <1%인 큰 구축물인 Cas9 플라스미드/RNA의 형질감염 방법을 우회한다. 본 Cas9-hiPSC 시스템은 인간 줄기 세포에서 고도로 효율적인 게놈 조작을 수행하기 위한 플랫폼으로서의 역할을 할 수 있다. 추가로, 피기백 시스템을 사용하여 hiPSC 내로 도입된 Cas9 카세트는 트랜스포사제 도입시 게놈으로부터 쉽게 제거될 수 있다.
실시예 XVII
포스파타제 처리된 가이드 RNA
Cas9-hiPSC에서의 연석적인 게놈 편집을 수행할 수 있도록 하기 위해, gRNA를 코딩하는 일련의 변형된 RNA를 생성하고, 리포솜과 함께 복합체로 Cas9-iPS 배양 배지 내로 보충되었다. 캡핑되지 않은, 포스파타제 처리된 천연 RNA은 앞서 보고된 5' 캡-Mod RNA보다 30X 더 큰 13%의 최적의 HDR 효율을 달성하였다 (도 16 참조).
한 측면에 따라, 가이드 RNA는 물리적으로 도너 DNA에 부착된다. 이러한 방식으로, 본 방법은 Cas9 매개 게놈 절단 및 ssODN-매개 HDR을 커플링시켜 서열 특이 게놈 편집을 자극하는 것을 제공한다. 최적의 농도로 DNA ssODN 도너와 연결된 gRNA는 44% HDR 및 비특이 NHEJ 2%를 달성하였다 (도 17 참조). 특히, 본 방법은 뉴클레오펙션 또는 전기천공시에 관찰되는 것과 같은 뚜렷한 독성을 초래하지 않는다.
한 측면에 따라, 본 개시내용은 게놈적으로 삽입된 Cas9와 함께 높은 형질감염 효율, 게놈 편집 효율을 달성한, gRNA를 코딩하는 시험관내 조작된 RNA 구조를 제공한다. 추가로, 본 개시내용은 상동성 지정 재조합 반응과 함께 게놈 절단 이벤트를 커플링하는 gRNA-DNA 키메라 구축물을 제공한다.
실시예 XVIII
가역적으로 조작된 Cas9를 hiPSC로부터 제거함으로써 흔적 없는 게놈 편집 달성
특정 측면에 따라, 가역성 벡터를 이용하여 Cas9 카세트를 hiPSC 세포의 게놈 내로 삽입한다. 따라서, 피기백 벡터를 이용함으로써 hiPSC 세포의 게놈 내로 Cas9 카세트를 가역적으로 삽입하였다. 세포를 트랜스포사제 코딩 플라스미드로 형질감염시킴으로써 게놈 편집된 hiPSC로부터 Cas9 카세트를 제거하였다. 따라서, 본 개시내용의 측면은 통상의 기술자에게 주지되어 있는, 가역성 벡터를 사용하는 것을 포함한다. 가역성 벡터는 게놈 내로 삽입될 수 있고, 이어서, 예를 들어, 상응하는 벡터 제거 효소로 제거될 수 있는 벡터이다. 상기 벡터 및 상응하는 벡터 제거 효소는 통상의 기술자에게 공지되어 있다. 전이증식된 iPS 세포에서 스크린을 수행하였고, PCR 반응에 의해 확인하여 Cas9-카세트가 없는 콜로니를 회수하였다. 따라서, 본 개시내용은 세포에 영구적인 Cas9 카세트가 존재하도록 함으로써 나머지 게놈에는 영향을 미치지 않으면서 게놈 편집을 수행할 수 있는 방법을 제공한다.
실시예 XIX
iPGP1 세포에서의 게놈 편집
심근증의 병인론에 관한 연구는 역사적으로 적합한 모델 시스템의 부족으로 인해 제대로 이루어지지 못했다. 환자 유래의 유도 다능성 줄기 세포 (iPSC)의 심장 근육 세포 분화는 이러한 장벽을 극복할 수 있는 한 유망한 방안을 제공하며, 심근증의 iPSC 모델링에 관한 보고가 출현하기 시작하였다. 그러나, 이러한 유망성을 실현하기 위해서는 환자 유래의 iPSC 세포주의 유전적 이형접합성을 극복할 수 있는 접근법을 필요로 할 것이다.
Cas9-iPGP1 세포주 및 DNA에 결합한 포스파타제 처리된 가이드 RNA를 사용하여, 바르트 증후군 환자에서는 단일 뉴클레오티드 결실을 보유하는 것으로 확인된 TAZ 엑손 6에서의 서열을 제외하면 동질유전자인 것인 3개의 iPSC 세포주를 생성하였다. 1회차의 RNA 형질감염으로 ~30%의 HDR 효율을 달성하였다. 원하는 돌연변이를 포함하는 변형된 Cas9-iPGP1 세포를 전이증식시키고 (도 18 참조), 세포주를 심장 근육 세포로 분화시켰다. 조작된 Cas9-iPGP1로부터 유래된 심장 근육 세포는 환자 유래의 iPSC에서, 및 신생 래트의 TAZ 넉다운 모델에서 관찰되는 카르디올리핀, 미토콘드리아, 및 ATP 결핍을 완전하게 재현하였다 (도 19 참조). 따라서, 다능성 세포에서 질환을 유발하는 돌연변이를 교정한 후, 세포를 원하는 세포 유형으로 분화시키는 방법을 제공한다.
실시예 XX
물질 및 방법
1. 피기백 Cas9 dox 유도성 안정한 인간 iPS/ES 세포주 확립.
1. 세포가 70% 컨플루언스에 도달한 후, 배양물을 최종 농도 10 uM의 ROCK 억제제 Y27632로 밤새도록 전처리한다.
2. 다음날, 멸균 1.5 ml 에펜도르프 튜브 중에서 82 ㎕의 인간 줄기 세포 뉴클레오펙터 용액 및 18 ul의 보충제 1을 조합하여 뉴클레오펙션 용액을 제조한다. 잘 혼합한다. 용액을 37℃에서 5 min 동안 인큐베이션시킨다.
3. mTeSR1을 흡인하고; 6 웰 플레이트 중 2 mL/웰로 DPBS를 이용하여 세포를 온화하게 세정한다.
4. DPBS를 흡인하고, 2 mL/웰의 베르센(Versene)을 첨가하고, 배양물이 한데 모이고, 느슨한 부착성을 띠되, 단, 탈착되지는 않을 때까지 배양물을 37℃ 인큐베이터에 다시 돌려 놓는다. 3-7 min이 소요된다.
5. 온화하게 베르센을 흡인하고, mTeSR1을 첨가한다. 1 ml mTeSR1을 첨가하고, 1,000 uL 마이크로피펫을 이용하여 세포 상으로 mTeSR1을 온화하게 유동시킴으로써 세포를 떼어낸다.
6. 떼어낸 세포를 수집하고, 그를 단일 세포 현탁액으로 온화하게 분쇄시키고, 혈구계수기에 의해 정량화하고, 세포 밀도를 1 ml당 세포 100만개인 밀도로 조정한다.
7. 1 ml 세포 현탁액을 1.5 ml 에펜도르프 튜브로 첨가하고, 벤치 탑 원심분리기에서 5 min 동안 1,100 RPM으로 원심분리시킨다.
8. 단계 2로부터의 100 ㎕ 인간 줄기 세포 뉴클레오펙터 용액 중에 세포를 재현탁시킨다.
9. 1 ml 피펫 팁을 이용하여 세포를 뉴클레오펙터 큐벳으로 옮겨 놓는다. 큐벳 중 세포 현탁액으로 1 ㎍의 플라스미드 트랜스포사제 및 5ug PB Cas9 플라스미드를 첨가한다. 온화하게 와동시켜 세포 및 DNA를 혼합한다.
10. 큐벳을 뉴클레오펙터 안으로 넣는다. 프로그램 B-016을 선택하고, X 버튼을 눌러 세포를 뉴클레오펙션시킨다.
11. 뉴클레오펙션시킨 후, 큐벳에 ROCK 억제제와 함께 500 ul mTeSR1 배지를 첨가한다.
12. 제공된 파스퇴르(Pasteur) 플라스틱 피펫을 이용하여 큐벳으로부터 뉴클레오펙션된 세포를 흡인한다. 이어서, ROCK 억제제와 함께 mTeSR1 배지가 있는 6 웰 플레이트의 마트리겔 코팅 웰 내로 세포를 적가 방식으로 옮겨 놓는다. 세포를 37℃에서 밤새도록 인큐베이션시킨다.
13. 다음날, 배지를 mTesr1로 교체하고, 72시간 동안의 형질감염 후; 1 ug/ml의 최종 농도로 퓨로마이신을 첨가한다. 이어서, 7일 이내에 세포주가 확립될 것이다.
2. RNA 제조
1. gRNA 코딩 서열 상류에 T7 프로모터를 포함하는 DNA 주형을 제조한다.
2.메가 클리어 퓨리피케이션(Mega Clear Purification)을 이용하여 DNA를 정제하고, 농도를 정규화시킨다.
3. 상이한 gRNA 제조를 위해 시판용 NTPS 혼합물을 제조한다.
4. 실온에서 시험관내 전사 믹스를 제조한다.
5. 37℃ (써모사이클러)에서 4시간 (3-6 hr도 허용) 동안 인큐베이션시킨다.
6.각 샘플에 2 ㎕ 터보(Turbo) DNAse (앰비온으로부터의 메가스크립트 키트)를 첨가한다. 온화하게 혼합하고, 37℃에서 15' 동안 인큐베이션시킨다.
7.제조사의 설명서에 따라 앰비온으로부터의 메가클리어를 이용하여 DNAse 처리된 반응물을 정제한다.
8. 메가클리어를 이용하여 RNA를 정제한다 (정제된 RNA를 수개월 동안 -80에서 보관할 수 있다).
9. 숙주 세포로부터의 톨(Toll) 2 면역 반응을 피하기 위해 포스페이트 기를 제거한다.
샘플을 온화하게 혼합하고, 37℃에서 30' (30'-1 hr도 허용) 동안 인큐베이션시킨다.
3. RNA 형질감염
1. 항생제 없이 48웰당 10K-20K 세포를 플레이팅한다. 형질감염을 위해 세포는 30-50% 컴플루언트여야 한다.
2. 형질감염 이전 적어도 2시간 전에 세포 배지를 B18R (200 ng/ml), DOX (1 ug/ml), 퓨로마이신 (2 ug/ml)을 포함하는 것으로 교체한다.
3. gRNA (0.5 ug~2 ug), 도너 DNA (0.5 ug~2 ug) 및 RNAiMax를 함유하는 형질감염 시약을 제조하고, 실온에서 15분 동안 혼합물을 인큐베이션시키고, 세포로 옮겨 놓는다.
4. 단일 인간 iPS 세포 시딩 및 단일클론 수집
1. 4일간의 dox 유도 및 1일간의 dox 제거 후, 배지를 흡인하고, DPBS를 이용하여 온화하게 세정한다. 2 ml/웰의 베르센을 첨가하고, 배양물을 그가 라운드 업되고, 분리되는 것이 아니라, 느슨하게 부착성을 띨 때까지 37℃ 인큐베이터에 다시 놓는다. 이를 통해 3-7 min이 소요된다.
2. 베르센을 온화하게 흡인하고, mTeSR1을 첨가한다. 1,000 uL 마이크로피펫을 이용하여 그 위로 mTeSR1을 온화하게 유동시킴으로써 1 ml mTeSR1을 첨가하고, 세포를 ?燦爭슈?.
3. 떼어낸 세포를 수집하고, 그를 단일 세포 현탁액으로 온화하게 연마하고, 혈구계수기에 의해 정량화하고, 세포 밀도를 100K 세포/ml로 조정한다.
4. 세포를 10 cm 디쉬당 50K, 100K 및 400K의 세포 밀도로 mTeSR1 + ROCK 억제제를 포함하는 마트리겔 코팅된 10 cm 디쉬 내로 시딩한다.
5. 단일 세포 형성 클론 스크리닝
1. 10 cm 디쉬에서 12일 동안 배양한 후, 클론은 육안으로 확인할 수 있고, 콜론 마커에 의해 표지화될 정도로 충분히 크다. 클론은 너무 크지 않게, 및 서로 부착되지 않게 한다.
2. 필터 팁이 있는 (10 ul로 설정된) P20 피펫을 사용하여 10 cm 디쉬를 배양 후드에 놓는다. 24 웰 플레이트의 한 웰에 대하여 10 ul 배지를 흡인한다. 클론을 작은 조각으로 스크래치하여 클론을 수집하고, 24 웰 플레이트 중 한 웰로 옮겨 놓는다. 각 클론에 대해 각각의 필터 팁을 이용한다.
3. 4-5일 경과 후, 24 웰 플레이트 중 한 웰 내의 클론의 크기는 분할할 수 있을 정도로 충분히 커진다.
4. 배지를 흡인하고, 2 mL/웰 DPBS로 세정한다.
5. DPBS를 흡인하고, 250 ul/웰 디스파제 (0.1 U/mL)로 교체하고, 37℃에서 7 min 동안 디스파제 중에서 세포를 인큐베이션시킨다.
6. 디스파제를 2 ml DPBS로 교체한다.
7. 250 ul mTeSR1을 첨가한다. 세포 스크랩퍼를 이용하여 세포를 떼어내고, 세포를 수집한다.
8. 125 ul 세포 현탁액을 마트리겔 코팅된 24 웰 플레이트의 웰로 옮겨 놓는다.
9. 게놈 DNA 추출을 위해 125 ul 세포 현탁액을 1.5 ml 에펜도르프 튜브로 옮겨 놓는다.
6. 클론 스크리닝
1. 단계 7.7로부터의 튜브를 원심분리한다.
2. 배지를 흡입하고, 웰당 250 ul 용해 완충제 (10 mM+트리스pH7.5+ (또는 +8.0), 10 mM EDTA,10 mM을 첨가한다.
3. NaCl, +10% SDS, 40 ug/mL +프로테이나제 K(완충제 사용 전 새로 첨가)를 첨가한다.
4. 55에서 밤새도록 인큐베이션시킨다.
5. 250 ul 이소프로판올을 첨가하여 DNA를 침전시킨다.
6. 고속으로 30분 동안 회전시킨다. 70% 에탄올로 세척한다.
7. 에탄올을 온화하게 제거한다. 5 min 동안 대기 건조시킨다.
8. 100-200 ul dH2O를 이용하여 gDNA를 재현탁시킨다.
9. 특이 프라이머를 이용하여 표적화된 게놈을 PCR 증폭시킨다.
10. 각 프라이머를 이용하여 PCR 생성물을 생어 서열분석한다.
11. 생어 서열 데이터를 분석하고, 표적화된 클론을 확장시킨다.
7. 피기백 벡터 제거
1. 단계 2.1-2.9를 반복한다.
2. 1 ml 피펫 팁을 이용하여 세포를 뉴클레오펙터 큐벳으로 옮겨 놓는다. 2 ㎍의 트랜스포사제 플라스미드를 큐벳 중의 세포 현탁액으로 첨가한다. 온화하게 와동시켜 세포 및 DNA를 혼합한다.
3. 단계 2.10-2.11을 반복한다.
4. 제공된 파스퇴르 플라스틱 피펫을 이용하여 큐벳으로부터 뉴클레오펙션된 세포를 흡입한다. 이어서, ROCK 억제제와 함께 mTeSR1 배지를 포함하는 10 cm 디쉬의 마트리겔 코팅된 웰 내로 세포를 적가 방식으로 옮겨 놓는다. 세포를 37℃에서 밤새도록 인큐베이션시켰다.
5. 다음날, 배지를 mTesr1로 교체하고, 다음 4일 동안 매일 배지를 교체한다.
6. 클론 크기가 충분히 크게 된 후, 20-50개의 클론을 선택하고, 24 웰에 시딩한다.
7. PB Cas9 피기백 벡터 프라이머를 이용하여 클론의 유전자형을 분석하고, 음석 클론을 확장시킨다.
참고 문헌
참고 문헌은 명세서 전역에 걸쳐 하기의 그의 번호로 명시되어 있으며, 마치 본원에 전체가 기술되어 있는 것과 같이 본 명세서에 내에 포함된다. 하기 참고 문헌들은 각각 그 전문이 본원에서 참조로 포함된다.
SEQUENCE LISTING
<110> Church, George
Yang, Luhan
Cargol, Marc
Yang, Joyce
<120> Genome Engineering
<130> 010498.00517
<140> PCT/US14/048140
<141> 2014-06-30
<150> US 61/858,866
<151> 2013-07-26
<160> 224
<170> PatentIn version 3.5
<210> 1
<211> 15
<212> DNA
<213> Artificial
<220>
<223> Oligonucleotide sequence
<400> 1
tggggcaagc ttctg 15
<210> 2
<211> 102
<212> DNA
<213> Artificial
<220>
<223> TALE template
<220>
<221> misc_feature
<222> (34)..(39)
<223> n is a, c, g, or t
<400> 2
ctaaccccag agcaggtcgt ggcaatcgcc tccnnnnnng gcggaaaaca ggcattggaa 60
acagtacagc ggctgctgcc ggtgctgtgc caagcgcacg ga 102
<210> 3
<211> 102
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<220>
<221> misc_feature
<222> (34)..(39)
<223> n is a, c, g, or t
<400> 3
ctaacccctg aacaggtagt cgctatagct tcannnnnng ggggcaagca agcacttgag 60
accgttcaac gactcctgcc agtgctctgc caagcccatg ga 102
<210> 4
<211> 102
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<220>
<221> misc_feature
<222> (34)..(39)
<223> n is a, c, g, or t
<400> 4
ttgactccgg agcaagtcgt cgcgatcgcg agcnnnnnng gggggaagca ggcgctggaa 60
actgttcaga gactgctgcc tgtactttgt caggcgcatg gt 102
<210> 5
<211> 102
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<220>
<221> misc_feature
<222> (34)..(39)
<223> n is a, c, g, or t
<400> 5
ctcacccccg aacaggttgt cgcaatagca agtnnnnnng gcggtaagca agccctagag 60
actgtgcaac gcctgctccc cgtgctgtgt caggctcacg gt 102
<210> 6
<211> 102
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<220>
<221> misc_feature
<222> (34)..(39)
<223> n is a, c, g, or t
<400> 6
ctgacacctg aacaagttgt cgcgatagcc agtnnnnnng ggggaaaaca agctctagaa 60
acggttcaaa ggttgttgcc cgttctgtgc caagcacatg gg 102
<210> 7
<211> 102
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<220>
<221> misc_feature
<222> (34)..(39)
<223> n is a, c, g, or t
<400> 7
ttaacacccg aacaagtagt agcgatagcg tcannnnnng ggggtaaaca ggctttggag 60
acggtacagc ggttattgcc ggtcctctgc caggcccacg ga 102
<210> 8
<211> 102
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<220>
<221> misc_feature
<222> (34)..(39)
<223> n is a, c, g, or t
<400> 8
cttacgccag aacaggtggt tgcaattgcc tccnnnnnng gcgggaaaca agcgttggaa 60
actgtgcaga gactccttcc tgttttgtgt caagcccacg gc 102
<210> 9
<211> 102
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<220>
<221> misc_feature
<222> (34)..(39)
<223> n is a, c, g, or t
<400> 9
ttgacgcctg agcaggttgt ggccatcgct agcnnnnnng gagggaagca ggctcttgaa 60
accgtacagc gacttctccc agttttgtgc caagctcacg gg 102
<210> 10
<211> 102
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<220>
<221> misc_feature
<222> (34)..(39)
<223> n is a, c, g, or t
<400> 10
ctaacccccg agcaagtagt tgccatagca agcnnnnnng gaggaaaaca ggcattagaa 60
acagttcagc gcttgctccc ggtactctgt caggcacacg gt 102
<210> 11
<211> 102
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<220>
<221> misc_feature
<222> (34)..(39)
<223> n is a, c, g, or t
<400> 11
ctaactccgg aacaggtcgt agccattgct tccnnnnnng gcggcaaaca ggcgctagag 60
acagtccaga ggctcttgcc tgtgttatgc caggcacatg gc 102
<210> 12
<211> 102
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<220>
<221> misc_feature
<222> (34)..(39)
<223> n is a, c, g, or t
<400> 12
ctcaccccgg agcaggtcgt tgccatcgcc agtnnnnnng gcggaaagca agctctcgaa 60
acagtacaac ggctgttgcc agtcctatgt caagctcatg ga 102
<210> 13
<211> 102
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<220>
<221> misc_feature
<222> (34)..(39)
<223> n is a, c, g, or t
<400> 13
ctgacgcccg agcaggtagt ggcaatcgca tctnnnnnng gaggtaaaca agcactcgag 60
actgtccaaa gattgttacc cgtactatgc caagcgcatg gt 102
<210> 14
<211> 102
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<220>
<221> misc_feature
<222> (34)..(39)
<223> n is a, c, g, or t
<400> 14
ttaaccccag agcaagttgt ggctattgca tctnnnnnng gtggcaaaca agccttggag 60
acagtgcaac gattactgcc tgtcttatgt caggcccatg gc 102
<210> 15
<211> 102
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<220>
<221> misc_feature
<222> (34)..(39)
<223> n is a, c, g, or t
<400> 15
cttactcctg agcaagtcgt agctatcgcc agcnnnnnng gtgggaaaca ggccctggaa 60
accgtacaac gtctcctccc agtactttgt caagcacacg gg 102
<210> 16
<211> 102
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<220>
<221> misc_feature
<222> (34)..(39)
<223> n is a, c, g, or t
<400> 16
ttgacaccgg aacaagtggt ggcgattgcg tccnnnnnng gaggcaagca ggcactggag 60
accgtccaac ggcttcttcc ggttctttgc caggctcatg gg 102
<210> 17
<211> 102
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<220>
<221> misc_feature
<222> (34)..(39)
<223> n is a, c, g, or t
<400> 17
ctcacgccag agcaggtggt agcaatagcg tcgnnnnnng gtggtaagca agcgcttgaa 60
acggtccagc gtcttctgcc ggtgttgtgc caggcgcacg ga 102
<210> 18
<211> 102
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<220>
<221> misc_feature
<222> (34)..(39)
<223> n is a, c, g, or t
<400> 18
ctcacaccag aacaagtggt tgctattgct agtnnnnnng gtggaaagca ggccctcgag 60
acggtgcaga ggttacttcc cgtcctctgt caagcgcacg gc 102
<210> 19
<211> 60
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<220>
<221> misc_feature
<222> (34)..(39)
<223> n is a, c, g, or t
<400> 19
ctcactccag agcaagtggt tgcgatcgct tcannnnnng gtggaagacc tgccctggaa 60
<210> 20
<211> 5714
<212> DNA
<213> Artificial
<220>
<223> Oligonucleotide sequence encoding TALEN backbone
<400> 20
atgtcgcgga cccggctccc ttccccaccc gcacccagcc cagcgttttc ggccgactcg 60
ttctcagacc tgcttaggca gttcgacccc tcactgttta acacatcgtt gttcgactcc 120
cttcctccgt ttggggcgca ccatacggag gcggccaccg gggagtggga tgaggtgcag 180
tcgggattga gagctgcgga tgcaccaccc ccaaccatgc gggtggccgt caccgctgcc 240
cgaccgccga gggcgaagcc cgcaccaagg cggagggcag cgcaaccgtc cgacgcaagc 300
cccgcagcgc aagtagattt gagaactttg ggatattcac agcagcagca ggaaaagatc 360
aagcccaaag tgaggtcgac agtcgcgcag catcacgaag cgctggtggg tcatgggttt 420
acacatgccc acatcgtagc cttgtcgcag caccctgcag cccttggcac ggtcgccgtc 480
aagtaccagg acatgattgc ggcgttgccg gaagccacac atgaggcgat cgtcggtgtg 540
gggaaacagt ggagcggagc ccgagcgctt gaggccctgt tgacggtcgc gggagagctg 600
agagggcctc cccttcagct ggacacgggc cagttgctga agatcgcgaa gcggggagga 660
gtcacggcgg tcgaggcggt gcacgcgtgg cgcaatgcgc tcacgggagc acccctcaac 720
agttcacgct gacagagacc gcggccgcat taggcacccc aggctttaca ctttatgctt 780
ccggctcgta taatgtgtgg attttgagtt aggatccgtc gagattttca ggagctaagg 840
aagctaaaat ggagaaaaaa atcactggat ataccaccgt tgatatatcc caatggcatc 900
gtaaagaaca ttttgaggca tttcagtcag ttgctcaatg tacctataac cagaccgttc 960
agctggatat tacggccttt ttaaagaccg taaagaaaaa taagcacaag ttttatccgg 1020
cctttattca cattcttgcc cgcctgatga atgctcatcc ggaattccgt atggcaatga 1080
aagacggtga gctggtgata tgggatagtg ttcacccttg ttacaccgtt ttccatgagc 1140
aaactgaaac gttttcatcg ctctggagtg aataccacga cgatttccgg cagtttctac 1200
acatatattc gcaagatgtg gcgtgttacg gtgaaaacct ggcctatttc cctaaagggt 1260
ttattgagaa tatgtttttc gtctcagcca atccctgggt gagtttcacc agttttgatt 1320
taaacgtggc caatatggac aacttcttcg cccccgtttt caccatgggc aaatattata 1380
cgcaaggcga caaggtgctg atgccgctgg cgattcaggt tcatcatgcc gtttgtgatg 1440
gcttccatgt cggcagaatg cttaatgaat tacaacagta ctgcgatgag tggcagggcg 1500
gggcgtaaag atctggatcc ggcttactaa aagccagata acagtatgcg tatttgcgcg 1560
ctgatttttg cggtataaga atatatactg atatgtatac ccgaagtatg tcaaaaagag 1620
gtatgctatg aagcagcgta ttacagtgac agttgacagc gacagctatc agttgctcaa 1680
ggcatatatg atgtcaatat ctccggtctg gtaagcacaa ccatgcagaa tgaagcccgt 1740
cgtctgcgtg ccgaacgctg gaaagcggaa aatcaggaag ggatggctga ggtcgcccgg 1800
tttattgaaa tgaacggctc ttttgctgac gagaacaggg gctggtgaaa tgcagtttaa 1860
ggtttacacc tataaaagag agagccgtta tcgtctgttt gtggatgtac agagtgatat 1920
tattgacacg cccgggcgac ggatggtgat ccccctggcc agtgcacgtc tgctgtcaga 1980
taaagtctcc cgtgaacttt acccggtggt gcatatcggg gatgaaagct ggcgcatgat 2040
gaccaccgat atggccagtg tgccggtctc cgttatcggg gaagaagtgg ctgatctcag 2100
ccaccgcgaa aatgacatca aaaacgccat taacctgatg ttctggggaa tataaatgtc 2160
aggctccctt atacacagcc agtctgcagg tcgacggtct cgctcttcga aggttacttc 2220
ccgtcctctg tcaagcgcac ggcctcactc cagagcaagt ggttgcgatc gcttcaaaca 2280
acggtggaag acctgccctg gaatcaatcg tggcccagct ttcgaggccg gaccccgcgc 2340
tggccgcact cactaatgat catcttgtag cgctggcctg cctcggcgga cgacccgcct 2400
tggatgcggt gaagaagggg ctcccgcacg cgcctgcatt gattaagcgg accaacagaa 2460
ggattcccga gaggacatca catcgagtgg caggttccca actcgtgaag agtgaacttg 2520
aggagaaaaa gtcggagctg cggcacaaat tgaaatacgt accgcatgaa tacatcgaac 2580
ttatcgaaat tgctaggaac tcgactcaag acagaatcct tgagatgaag gtaatggagt 2640
tctttatgaa ggtttatgga taccgaggga agcatctcgg tggatcacga aaacccgacg 2700
gagcaatcta tacggtgggg agcccgattg attacggagt gatcgtcgac acgaaagcct 2760
acagcggtgg gtacaatctt cccatcgggc aggcagatga gatgcaacgt tatgtcgaag 2820
aaaatcagac caggaacaaa cacatcaatc caaatgagtg gtggaaagtg tatccttcat 2880
cagtgaccga gtttaagttt ttgtttgtct ctgggcattt caaaggcaac tataaggccc 2940
agctcacacg gttgaatcac attacgaact gcaatggtgc ggttttgtcc gtagaggaac 3000
tgctcattgg tggagaaatg atcaaagcgg gaactctgac actggaagaa gtcagacgca 3060
agtttaacaa tggcgagatc aatttccgca agctaaaatg gagaaaaaaa tcactggata 3120
taccaccgtt gatatatccc aatggcatcg taaagaacat tttgaggcat ttcagtcagt 3180
tgctcaatgt acctataacc agaccgttca gctggatatt acggcctttt taaagaccgt 3240
aaagaaaaat aagcacaagt tttatccggc ctttattcac attcttgccc gcctgatgaa 3300
tgctcatccg gaattccgta tggcaatgaa agacggtgag ctggtgatat gggatagtgt 3360
tcacccttgt tacaccgttt tccatgagca aactgaaacg ttttcatcgc tctggagtga 3420
ataccacgac gatttccggc agtttctaca catatattcg caagatgtgg cgtgttacgg 3480
tgaaaacctg gcctatttcc ctaaagggtt tattgagaat atgtttttcg tctcagccaa 3540
tccctgggtg agtttcacca gttttgattt aaacgtggcc aatatggaca acttcttcgc 3600
ccccgttttc accatgggca aatattatac gcaaggcgac aaggtgctga tgccgctggc 3660
gattcaggtt catcatgccg tttgtgatgg cttccatgtc ggcagaatgc ttaatgaatt 3720
acaacagtac tgcgatgagt ggcagggcgg ggcgtaaaga tctggatccg gcttactaaa 3780
agccagataa cagtatgcgt atttgcgcgc tgatttttgc ggtataagaa tatatactga 3840
tatgtatacc cgaagtatgt caaaaagagg tatgctatga agcagcgtat tacagtgaca 3900
gttgacagcg acagctatca gttgctcaag gcatatatga tgtcaatatc tccggtctgg 3960
taagcacaac catgcagaat gaagcccgtc gtctgcgtgc cgaacgctgg aaagcggaaa 4020
atcaggaagg gatggctgag gtcgcccggt ttattgaaat gaacggctct tttgctgacg 4080
agaacagggg ctggtgaaat gcagtttaag gtttacacct ataaaagaga gagccgttat 4140
cgtctgtttg tggatgtaca gagtgatatt attgacacgc ccgggcgacg gatggtgatc 4200
cccctggcca gtgcacgtct gctgtcagat aaagtctccc gtgaacttta cccggtggtg 4260
catatcgggg atgaaagctg gcgcatgatg accaccgata tggccagtgt gccggtctcc 4320
gttatcgggg aagaagtggc tgatctcagc caccgcgaaa atgacatcaa aaacgccatt 4380
aacctgatgt tctggggaat ataaatgtca ggctccctta tacacagcca gtctgcaggt 4440
cgacggtctc gctcttcgaa ggttacttcc cgtcctctgt caagcgcacg gcctcactcc 4500
agagcaagtg gttgcgatcg cttcaaacaa cggtggaaga cctgccctgg aatcaatcgt 4560
ggcccagctt tcgaggccgg accccgcgct ggccgcactc actaatgatc atcttgtagc 4620
gctggcctgc ctcggcggac gacccgcctt ggatgcggtg aagaaggggc tcccgcacgc 4680
gcctgcattg attaagcgga ccaacagaag gattcccgag aggacatagc cccaagaaga 4740
agagaaaggt ggaggccagc ggttccggac gggctgacgc attggacgat tttgatctgg 4800
atatgctggg aagtgacgcc ctcgatgatt ttgaccttga catgcttggt tcggatgccc 4860
ttgatgactt tgacctcgac atgctcggca gtgacgccct tgatgatttc gacctggaca 4920
tgctgattaa ctctagaggc agtggagagg gcagaggaag tctgctaaca tgcggtgacg 4980
tcgaggagaa tcctggccca gtgagcaagg gcgaggagct gttcaccggg gtggtgccca 5040
tcctggtcga gctggacggc gacgtaaacg gccacaagtt cagcgtgtcc ggcgagggcg 5100
agggcgatgc cacctacggc aagctgaccc tgaagttcat ctgcaccacc ggcaagctgc 5160
ccgtgccctg gcccaccctc gtgaccaccc tgacctacgg cgtgcagtgc ttcagccgct 5220
accccgacca catgaagcag cacgacttct tcaagtccgc catgcccgaa ggctacgtcc 5280
aggagcgcac catcttcttc aaggacgacg gcaactacaa gacccgcgcc gaggtgaagt 5340
tcgagggcga caccctggtg aaccgcatcg agctgaaggg catcgacttc aaggaggacg 5400
gcaacatcct ggggcacaag ctggagtaca actacaacag ccacaacgtc tatatcatgg 5460
ccgacaagca gaagaacggc atcaaggtga acttcaagat ccgccacaac atcgaggacg 5520
gcagcgtgca gctcgccgac cactaccagc agaacacccc catcggcgac ggccccgtgc 5580
tgctgcccga caaccactac ctgagcaccc agtccgccct gagcaaagac cccaacgaga 5640
agcgcgatca catggtcctg ctggagttcg tgaccgccgc cgggatcact ctcggcatgg 5700
acgagctgta caag 5714
<210> 21
<211> 3465
<212> DNA
<213> Artificial
<220>
<223> Oligonucleotide sequence encoding TALE backbone sequence
<400> 21
atgtcgcgga cccggctccc ttccccaccc gcacccagcc cagcgttttc ggccgactcg 60
ttctcagacc tgcttaggca gttcgacccc tcactgttta acacatcgtt gttcgactcc 120
cttcctccgt ttggggcgca ccatacggag gcggccaccg gggagtggga tgaggtgcag 180
tcgggattga gagctgcgga tgcaccaccc ccaaccatgc gggtggccgt caccgctgcc 240
cgaccgccga gggcgaagcc cgcaccaagg cggagggcag cgcaaccgtc cgacgcaagc 300
cccgcagcgc aagtagattt gagaactttg ggatattcac agcagcagca ggaaaagatc 360
aagcccaaag tgaggtcgac agtcgcgcag catcacgaag cgctggtggg tcatgggttt 420
acacatgccc acatcgtagc cttgtcgcag caccctgcag cccttggcac ggtcgccgtc 480
aagtaccagg acatgattgc ggcgttgccg gaagccacac atgaggcgat cgtcggtgtg 540
gggaaacagt ggagcggagc ccgagcgctt gaggccctgt tgacggtcgc gggagagctg 600
agagggcctc cccttcagct ggacacgggc cagttgctga agatcgcgaa gcggggagga 660
gtcacggcgg tcgaggcggt gcacgcgtgg cgcaatgcgc tcacgggagc acccctcaac 720
agttcacgct gacagagacc gcggccgcat taggcacccc aggctttaca ctttatgctt 780
ccggctcgta taatgtgtgg attttgagtt aggatccgtc gagattttca ggagctaagg 840
aagctaaaat ggagaaaaaa atcactggat ataccaccgt tgatatatcc caatggcatc 900
gtaaagaaca ttttgaggca tttcagtcag ttgctcaatg tacctataac cagaccgttc 960
agctggatat tacggccttt ttaaagaccg taaagaaaaa taagcacaag ttttatccgg 1020
cctttattca cattcttgcc cgcctgatga atgctcatcc ggaattccgt atggcaatga 1080
aagacggtga gctggtgata tgggatagtg ttcacccttg ttacaccgtt ttccatgagc 1140
aaactgaaac gttttcatcg ctctggagtg aataccacga cgatttccgg cagtttctac 1200
acatatattc gcaagatgtg gcgtgttacg gtgaaaacct ggcctatttc cctaaagggt 1260
ttattgagaa tatgtttttc gtctcagcca atccctgggt gagtttcacc agttttgatt 1320
taaacgtggc caatatggac aacttcttcg cccccgtttt caccatgggc aaatattata 1380
cgcaaggcga caaggtgctg atgccgctgg cgattcaggt tcatcatgcc gtttgtgatg 1440
gcttccatgt cggcagaatg cttaatgaat tacaacagta ctgcgatgag tggcagggcg 1500
gggcgtaaag atctggatcc ggcttactaa aagccagata acagtatgcg tatttgcgcg 1560
ctgatttttg cggtataaga atatatactg atatgtatac ccgaagtatg tcaaaaagag 1620
gtatgctatg aagcagcgta ttacagtgac agttgacagc gacagctatc agttgctcaa 1680
ggcatatatg atgtcaatat ctccggtctg gtaagcacaa ccatgcagaa tgaagcccgt 1740
cgtctgcgtg ccgaacgctg gaaagcggaa aatcaggaag ggatggctga ggtcgcccgg 1800
tttattgaaa tgaacggctc ttttgctgac gagaacaggg gctggtgaaa tgcagtttaa 1860
ggtttacacc tataaaagag agagccgtta tcgtctgttt gtggatgtac agagtgatat 1920
tattgacacg cccgggcgac ggatggtgat ccccctggcc agtgcacgtc tgctgtcaga 1980
taaagtctcc cgtgaacttt acccggtggt gcatatcggg gatgaaagct ggcgcatgat 2040
gaccaccgat atggccagtg tgccggtctc cgttatcggg gaagaagtgg ctgatctcag 2100
ccaccgcgaa aatgacatca aaaacgccat taacctgatg ttctggggaa tataaatgtc 2160
aggctccctt atacacagcc agtctgcagg tcgacggtct cgctcttcga aggttacttc 2220
ccgtcctctg tcaagcgcac ggcctcactc cagagcaagt ggttgcgatc gcttcaaaca 2280
acggtggaag acctgccctg gaatcaatcg tggcccagct ttcgaggccg gaccccgcgc 2340
tggccgcact cactaatgat catcttgtag cgctggcctg cctcggcgga cgacccgcct 2400
tggatgcggt gaagaagggg ctcccgcacg cgcctgcatt gattaagcgg accaacagaa 2460
ggattcccga gaggacatag ccccaagaag aagagaaagg tggaggccag cggttccgga 2520
cgggctgacg cattggacga ttttgatctg gatatgctgg gaagtgacgc cctcgatgat 2580
tttgaccttg acatgcttgg ttcggatgcc cttgatgact ttgacctcga catgctcggc 2640
agtgacgccc ttgatgattt cgacctggac atgctgatta actctagagg cagtggagag 2700
ggcagaggaa gtctgctaac atgcggtgac gtcgaggaga atcctggccc agtgagcaag 2760
ggcgaggagc tgttcaccgg ggtggtgccc atcctggtcg agctggacgg cgacgtaaac 2820
ggccacaagt tcagcgtgtc cggcgagggc gagggcgatg ccacctacgg caagctgacc 2880
ctgaagttca tctgcaccac cggcaagctg cccgtgccct ggcccaccct cgtgaccacc 2940
ctgacctacg gcgtgcagtg cttcagccgc taccccgacc acatgaagca gcacgacttc 3000
ttcaagtccg ccatgcccga aggctacgtc caggagcgca ccatcttctt caaggacgac 3060
ggcaactaca agacccgcgc cgaggtgaag ttcgagggcg acaccctggt gaaccgcatc 3120
gagctgaagg gcatcgactt caaggaggac ggcaacatcc tggggcacaa gctggagtac 3180
aactacaaca gccacaacgt ctatatcatg gccgacaagc agaagaacgg catcaaggtg 3240
aacttcaaga tccgccacaa catcgaggac ggcagcgtgc agctcgccga ccactaccag 3300
cagaacaccc ccatcggcga cggccccgtg ctgctgcccg acaaccacta cctgagcacc 3360
cagtccgccc tgagcaaaga ccccaacgag aagcgcgatc acatggtcct gctggagttc 3420
gtgaccgccg ccgggatcac tctcggcatg gacgagctgt acaag 3465
<210> 22
<211> 1368
<212> PRT
<213> Streptococcus pyogenes
<400> 22
Met Asp Lys Lys Tyr Ser Ile Gly Leu Asp Ile Gly Thr Asn Ser Val
1 5 10 15
Gly Trp Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe
20 25 30
Lys Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile
35 40 45
Gly Ala Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu
50 55 60
Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys
65 70 75 80
Tyr Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser
85 90 95
Phe Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys
100 105 110
His Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr
115 120 125
His Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp
130 135 140
Ser Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His
145 150 155 160
Met Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro
165 170 175
Asp Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr
180 185 190
Asn Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala
195 200 205
Lys Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn
210 215 220
Leu Ile Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn
225 230 235 240
Leu Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe
245 250 255
Asp Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp
260 265 270
Asp Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp
275 280 285
Leu Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp
290 295 300
Ile Leu Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser
305 310 315 320
Met Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys
325 330 335
Ala Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe
340 345 350
Asp Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser
355 360 365
Gln Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp
370 375 380
Gly Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg
385 390 395 400
Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu
405 410 415
Gly Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe
420 425 430
Leu Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile
435 440 445
Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp
450 455 460
Met Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu
465 470 475 480
Val Val Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr
485 490 495
Asn Phe Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser
500 505 510
Leu Leu Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys
515 520 525
Tyr Val Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln
530 535 540
Lys Lys Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr
545 550 555 560
Val Lys Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp
565 570 575
Ser Val Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly
580 585 590
Thr Tyr His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp
595 600 605
Asn Glu Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr
610 615 620
Leu Phe Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala
625 630 635 640
His Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr
645 650 655
Thr Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp
660 665 670
Lys Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe
675 680 685
Ala Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe
690 695 700
Lys Glu Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu
705 710 715 720
His Glu His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly
725 730 735
Ile Leu Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly
740 745 750
Arg His Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln
755 760 765
Thr Thr Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile
770 775 780
Glu Glu Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro
785 790 795 800
Val Glu Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu
805 810 815
Gln Asn Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg
820 825 830
Leu Ser Asp Tyr Asp Val Asp His Ile Val Pro Gln Ser Phe Leu Lys
835 840 845
Asp Asp Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg
850 855 860
Gly Lys Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys
865 870 875 880
Asn Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys
885 890 895
Phe Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp
900 905 910
Lys Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr
915 920 925
Lys His Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp
930 935 940
Glu Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
945 950 955 960
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg
965 970 975
Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val
980 985 990
Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe
995 1000 1005
Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala
1010 1015 1020
Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe
1025 1030 1035
Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala
1040 1045 1050
Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu
1055 1060 1065
Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val
1070 1075 1080
Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys Thr
1085 1090 1095
Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro Lys
1100 1105 1110
Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp Pro
1115 1120 1125
Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala Tyr Ser Val
1130 1135 1140
Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys Lys Leu Lys
1145 1150 1155
Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu Arg Ser Ser
1160 1165 1170
Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys Gly Tyr Lys
1175 1180 1185
Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr Ser Leu
1190 1195 1200
Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala Gly
1205 1210 1215
Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr Val
1220 1225 1230
Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser
1235 1240 1245
Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys
1250 1255 1260
His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys
1265 1270 1275
Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala
1280 1285 1290
Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn
1295 1300 1305
Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala
1310 1315 1320
Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr Thr Ser
1325 1330 1335
Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln Ser Ile Thr
1340 1345 1350
Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly Gly Asp
1355 1360 1365
<210> 23
<211> 82
<212> DNA
<213> Artificial
<220>
<223> Oligonucleotide sequence
<220>
<221> misc_feature
<222> (41)..(41)
<223> n is a, c, g, or t
<400> 23
ttcttggctt tatatatctt gtggaaagga cgaaacaccg ngttttagag ctagaaatag 60
caagttaaaa taaggctagt cc 82
<210> 24
<211> 1692
<212> DNA
<213> Artificial
<220>
<223> Oligonucleotide sequence encoding re-TALE
<400> 24
ctaacccctg aacaggtagt cgctatagct tcaaatatcg ggggcaagca agcacttgag 60
accgttcaac gactcctgcc agtgctctgc caagcccatg gattgactcc ggagcaagtc 120
gtcgcgatcg cgagcaacgg cggggggaag caggcgctgg aaactgttca gagactgctg 180
cctgtacttt gtcaggcgca tggtctcacc cccgaacagg ttgtcgcaat agcaagtaat 240
ataggcggta agcaagccct agagactgtg caacgcctgc tccccgtgct gtgtcaggct 300
cacggtctga cacctgaaca agttgtcgcg atagccagtc acgacggggg aaaacaagct 360
ctagaaacgg ttcaaaggtt gttgcccgtt ctgtgccaag cacatgggtt aacacccgaa 420
caagtagtag cgatagcgtc aaataacggg ggtaaacagg ctttggagac ggtacagcgg 480
ttattgccgg tcctctgcca ggcccacgga cttacgccag aacaggtggt tgcaattgcc 540
tccaacatcg gcgggaaaca agcgttggaa actgtgcaga gactccttcc tgttttgtgt 600
caagcccacg gcttgacgcc tgagcaggtt gtggccatcg ctagccacga cggagggaag 660
caggctcttg aaaccgtaca gcgacttctc ccagttttgt gccaagctca cgggctaacc 720
cccgagcaag tagttgccat agcaagcaac ggaggaggaa aacaggcatt agaaacagtt 780
cagcgcttgc tcccggtact ctgtcaggca cacggtctaa ctccggaaca ggtcgtagcc 840
attgcttccc atgatggcgg caaacaggcg ctagagacag tccagaggct cttgcctgtg 900
ttatgccagg cacatggcct caccccggag caggtcgttg ccatcgccag taatatcggc 960
ggaaagcaag ctctcgaaac agtacaacgg ctgttgccag tcctatgtca agctcatgga 1020
ctgacgcccg agcaggtagt ggcaatcgca tctcacgatg gaggtaaaca agcactcgag 1080
actgtccaaa gattgttacc cgtactatgc caagcgcatg gtttaacccc agagcaagtt 1140
gtggctattg catctaacgg cggtggcaaa caagccttgg agacagtgca acgattactg 1200
cctgtcttat gtcaggccca tggccttact cctgagcaag tcgtagctat cgccagcaac 1260
ataggtggga aacaggccct ggaaaccgta caacgtctcc tcccagtact ttgtcaagca 1320
cacgggttga caccggaaca agtggtggcg attgcgtcca acggcggagg caagcaggca 1380
ctggagaccg tccaacggct tcttccggtt ctttgccagg ctcatgggct cacgccagag 1440
caggtggtag caatagcgtc gaacatcggt ggtaagcaag cgcttgaaac ggtccagcgt 1500
cttctgccgg tgttgtgcca ggcgcacgga ctcacaccag aacaagtggt tgctattgct 1560
agtaacaacg gtggaaagca ggccctcgag acggtgcaga ggttacttcc cgtcctctgt 1620
caagcgcacg gcctcactcc agagcaagtg gttgcgatcg cttcaaacaa tggtggaaga 1680
cctgccctgg aa 1692
<210> 25
<211> 237
<212> DNA
<213> Artificial
<220>
<223> Template sequence
<220>
<221> misc_feature
<222> (64)..(69)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (166)..(171)
<223> n is a, c, g, or t
<400> 25
cgcaatgcgc tcacgggagc acccctcaac ctaacccctg aacaggtagt cgctatagct 60
tcannnnnng ggggcaagca agcacttgag accgttcaac gactcctgcc agtgctctgc 120
caagcccatg gattgactcc ggagcaagtc gtcgcgatcg cgagcnnnnn nggggggaag 180
caggcgctgg aaactgttca gagactgctg cctgtacttt gtcaggcgca tggtctc 237
<210> 26
<211> 240
<212> DNA
<213> Artificial
<220>
<223> Template sequence
<220>
<221> misc_feature
<222> (67)..(72)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (169)..(174)
<223> n is a, c, g, or t
<400> 26
agactgctgc ctgtactttg tcaggcgcat ggtctcaccc ccgaacaggt tgtcgcaata 60
gcaagtnnnn nnggcggtaa gcaagcccta gagactgtgc aacgcctgct ccccgtgctg 120
tgtcaggctc acggtctgac acctgaacaa gttgtcgcga tagccagtnn nnnnggggga 180
aaacaagctc tagaaacggt tcaaaggttg ttgcccgttc tgtgccaagc acatgggtta 240
<210> 27
<211> 232
<212> DNA
<213> Artificial
<220>
<223> Template sequence
<220>
<221> misc_feature
<222> (59)..(64)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (161)..(166)
<223> n is a, c, g, or t
<400> 27
tgcgctcacg ggagcacccc tcaacctcac ccccgaacag gttgtcgcaa tagcaagtnn 60
nnnnggcggt aagcaagccc tagagactgt gcaacgcctg ctccccgtgc tgtgtcaggc 120
tcacggtctg acacctgaac aagttgtcgc gatagccagt nnnnnngggg gaaaacaagc 180
tctagaaacg gttcaaaggt tgttgcccgt tctgtgccaa gcacatgggt ta 232
<210> 28
<211> 246
<212> DNA
<213> Artificial
<220>
<223> Template sequence
<220>
<221> misc_feature
<222> (67)..(72)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (169)..(174)
<223> n is a, c, g, or t
<400> 28
aggttgttgc ccgttctgtg ccaagcacat gggttaacac ccgaacaagt agtagcgata 60
gcgtcannnn nngggggtaa acaggctttg gagacggtac agcggttatt gccggtcctc 120
tgccaggccc acggacttac gccagaacag gtggttgcaa ttgcctccnn nnnnggcggg 180
aaacaagcgt tggaaactgt gcagagactc cttcctgttt tgtgtcaagc ccacggcttg 240
acgcct 246
<210> 29
<211> 240
<212> DNA
<213> Artificial
<220>
<223> Template sequence
<220>
<221> misc_feature
<222> (67)..(72)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (169)..(174)
<223> n is a, c, g, or t
<400> 29
agactccttc ctgttttgtg tcaagcccac ggcttgacgc ctgagcaggt tgtggccatc 60
gctagcnnnn nnggagggaa gcaggctctt gaaaccgtac agcgacttct cccagttttg 120
tgccaagctc acgggctaac ccccgagcaa gtagttgcca tagcaagcnn nnnnggagga 180
aaacaggcat tagaaacagt tcagcgcttg ctcccggtac tctgtcaggc acacggtcta 240
<210> 30
<211> 240
<212> DNA
<213> Artificial
<220>
<223> Template sequence
<220>
<221> misc_feature
<222> (67)..(72)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (169)..(174)
<223> n is a, c, g, or t
<400> 30
cgcttgctcc cggtactctg tcaggcacac ggtctaactc cggaacaggt cgtagccatt 60
gcttccnnnn nnggcggcaa acaggcgcta gagaccgtcc agaggctctt gcctgtgtta 120
tgccaggcac atggcctcac cccggagcag gtcgttgcca tcgccagtnn nnnnggcgga 180
aagcaagctc tcgaaacagt acaacggctg ttgccagtcc tatgtcaagc tcatggactg 240
<210> 31
<211> 240
<212> DNA
<213> Artificial
<220>
<223> Template sequence
<220>
<221> misc_feature
<222> (67)..(72)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (169)..(174)
<223> n is a, c, g, or t
<400> 31
cggctgttgc cagtcctatg tcaagctcat ggactgacgc ccgagcaggt agtggcaatc 60
gcatctnnnn nnggaggtaa acaagcactc gagactgtcc aaagattgtt acccgtacta 120
tgccaagcgc atggtttaac cccagagcaa gttgtggcta ttgcatctnn nnnnggtggc 180
aaacaagcct tggagaccgt gcaacgatta ctgcctgtct tatgtcaggc ccatggcctt 240
<210> 32
<211> 240
<212> DNA
<213> Artificial
<220>
<223> Template sequence
<220>
<221> misc_feature
<222> (67)..(72)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (169)..(174)
<223> n is a, c, g, or t
<400> 32
cgattactgc ctgtcttatg tcaggcccat ggccttactc ctgagcaggt ggtcgctatc 60
gccagcnnnn nngggggcaa gcaagcactg gaaacagtcc agcgtttgct tccagtactt 120
tgtcaggcgc atggattgac accggaacaa gtggtggcta tagcctcann nnnnggagga 180
aagcaggcgc tggaaaccgt ccaacgtctt ttaccggtgc tttgccaggc gcacgggctc 240
<210> 33
<211> 240
<212> DNA
<213> Artificial
<220>
<223> Template sequence
<220>
<221> misc_feature
<222> (67)..(72)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (169)..(174)
<223> n is a, c, g, or t
<400> 33
cgattactgc ctgtcttatg tcaggcccat ggccttactc ctgagcaagt cgtagctatc 60
gccagcnnnn nnggtgggaa acaggccctg gaaaccgtac aacgtctcct cccagtactt 120
tgtcaagcac acgggttgac accggaacaa gtggtggcga ttgcgtccnn nnnnggaggc 180
aagcaggcac tggagaccgt ccaacggctt cttccggttc tttgccaggc tcatgggctc 240
<210> 34
<211> 240
<212> DNA
<213> Artificial
<220>
<223> Template sequence
<220>
<221> misc_feature
<222> (67)..(72)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (169)..(174)
<223> n is a, c, g, or t
<400> 34
cggcttcttc cggttctttg ccaggctcat gggctcacgc cagagcaggt ggtagcaata 60
gcgtcgnnnn nnggtggtaa gcaagcgctt gaaacggtcc agcgtcttct gccggtgttg 120
tgccaggcgc acggactcac accagaacaa gtggttgcta ttgctagtnn nnnnggtgga 180
aagcaggccc tcgagacggt gcagaggtta cttcccgtcc tctgtcaagc gcacggcctc 240
<210> 35
<211> 49
<212> DNA
<213> Artificial
<220>
<223> Primer sequence
<400> 35
cgcaatgcgc tcacgggagc acccctcaac ctaacccctg aacaggtag 49
<210> 36
<211> 46
<212> DNA
<213> Artificial
<220>
<223> Primer sequence
<400> 36
gagaccatgc gcctgacaaa gtacaggcag cagtctctga acagtt 46
<210> 37
<211> 33
<212> DNA
<213> Artificial
<220>
<223> Primer sequence
<400> 37
tggcgcaatg cgctcacggg agcacccctc aac 33
<210> 38
<211> 49
<212> DNA
<213> Artificial
<220>
<223> Primer sequence
<400> 38
agactgctgc ctgtactttg tcaggcgcat ggtctcaccc ccgaacagg 49
<210> 39
<211> 46
<212> DNA
<213> Artificial
<220>
<223> Primer sequence
<400> 39
taacccatgt gcttggcaca gaacgggcaa caacctttga accgtt 46
<210> 40
<211> 48
<212> DNA
<213> Artificial
<220>
<223> Primer sequence
<400> 40
aggttgttgc ccgttctgtg ccaagcacat gggttaacac ccgaacaa 48
<210> 41
<211> 52
<212> DNA
<213> Artificial
<220>
<223> Primer sequence
<400> 41
aggcgtcaag ccgtgggctt gacacaaaac aggaaggagt ctctgcacag tt 52
<210> 42
<211> 45
<212> DNA
<213> Artificial
<220>
<223> Primer sequence
<400> 42
agactccttc ctgttttgtg tcaagcccac ggcttgacgc ctgag 45
<210> 43
<211> 40
<212> DNA
<213> Artificial
<220>
<223> Primer sequence
<400> 43
tagaccgtgt gcctgacaga gtaccgggag caagcgctga 40
<210> 44
<211> 39
<212> DNA
<213> Artificial
<220>
<223> Primer sequence
<400> 44
cgcttgctcc cggtactctg tcaggcacac ggtctaact 39
<210> 45
<211> 40
<212> DNA
<213> Artificial
<220>
<223> Primer sequence
<400> 45
cagtccatga gcttgacata ggactggcaa cagccgttgt 40
<210> 46
<211> 39
<212> DNA
<213> Artificial
<220>
<223> Primer sequence
<400> 46
cggctgttgc cagtcctatg tcaagctcat ggactgacg 39
<210> 47
<211> 40
<212> DNA
<213> Artificial
<220>
<223> Primer sequence
<400> 47
aaggccatgg gcctgacata agacaggcag taatcgttgc 40
<210> 48
<211> 39
<212> DNA
<213> Artificial
<220>
<223> Primer sequence
<400> 48
cgattactgc ctgtcttatg tcaggcccat ggccttact 39
<210> 49
<211> 43
<212> DNA
<213> Artificial
<220>
<223> Primer sequence
<400> 49
gagcccgtgc gcctggcaaa gcaccggtaa aagacgttgg acg 43
<210> 50
<211> 50
<212> DNA
<213> Artificial
<220>
<223> Primer sequence
<400> 50
cgattactgc ctgtcttatg tcaggcccat ggccttactc ctgagcaagt 50
<210> 51
<211> 40
<212> DNA
<213> Artificial
<220>
<223> Primer sequence
<400> 51
gagcccatga gcctggcaaa gaaccggaag aagccgttgg 40
<210> 52
<211> 51
<212> DNA
<213> Artificial
<220>
<223> Primer sequence
<400> 52
cggcttcttc cggttctttg ccaggctcat gggctcacgc cagagcaggt g 51
<210> 53
<211> 40
<212> DNA
<213> Artificial
<220>
<223> Primer sequence
<400> 53
gaggccgtgc gcttgacaga ggacgggaag taacctctgc 40
<210> 54
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 54
tccccacttt cttgtgaa 18
<210> 55
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 55
taaccactca ggacaggg 18
<210> 56
<211> 19
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 56
cactttcttg tgaatcctt 19
<210> 57
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 57
tcacacagca agtcagca 18
<210> 58
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 58
tagcggagca ggctcgga 18
<210> 59
<211> 19
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 59
tgggctagcg gagcaggct 19
<210> 60
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 60
tacccagacg agaaagct 18
<210> 61
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 61
tcagactgcc aagcttga 18
<210> 62
<211> 19
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 62
acccagacga gaaagctga 19
<210> 63
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 63
tcttgtggct cgggagta 18
<210> 64
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 64
tattgtcagc agagctga 18
<210> 65
<211> 19
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 65
agagggcatc ttgtggctc 19
<210> 66
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 66
ttgagatttt cagatgtc 18
<210> 67
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 67
tatacagtca tatcaagc 18
<210> 68
<211> 19
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 68
atcaagctct cttggcggt 19
<210> 69
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 69
ttcagataga ttatatct 18
<210> 70
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 70
tgccagatac ataggtgg 18
<210> 71
<211> 19
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 71
gcttcagata gattatatc 19
<210> 72
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 72
ttatactgtc tatatgat 18
<210> 73
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 73
tcagctcttc tggccaga 18
<210> 74
<211> 19
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 74
acggatgtct cagctcttc 19
<210> 75
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 75
tggccagaag agctgaga 18
<210> 76
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 76
ttaccgggga gagtttct 18
<210> 77
<211> 19
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 77
ccggggagag tttcttgta 19
<210> 78
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 78
tttgcagaga gatgagtc 18
<210> 79
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 79
ttagcagaag ataagatt 18
<210> 80
<211> 19
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 80
gaaatcttat cttctgcta 19
<210> 81
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 81
tataagacta aactaccc 18
<210> 82
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 82
tcgtctgcca ccacagat 18
<210> 83
<211> 19
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 83
aatgcatgac attcatctg 19
<210> 84
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 84
taaaacagtt tgcattca 18
<210> 85
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 85
tataaagtcc tagaatgt 18
<210> 86
<211> 19
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 86
aacagtttgc attcatgga 19
<210> 87
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 87
tggccatctc tgacctgt 18
<210> 88
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 88
tagtgagccc agaagggg 18
<210> 89
<211> 19
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 89
ccagaagggg acagtaaga 19
<210> 90
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 90
taggtacctg gctgtcgt 18
<210> 91
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 91
tgaccgtcct ggctttta 18
<210> 92
<211> 19
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 92
ctgacaatcg ataggtacc 19
<210> 93
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 93
tgtcatggtc atctgcta 18
<210> 94
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 94
tcgacaccga agcagagt 18
<210> 95
<211> 19
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 95
acaccgaagc agagttttt 19
<210> 96
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 96
tgcccccgcg aggccaca 18
<210> 97
<211> 18
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 97
tctggaagtt gaacaccc 18
<210> 98
<211> 19
<212> DNA
<213> Artificial
<220>
<223> Targeting sequence
<400> 98
ggaagttgaa cacccttgc 19
<210> 99
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 99
ctactgtcat tcagggcaat acccagacga gaaagctgag ggtataacag gtttcaagct 60
tggcagtctg actacagagg ccactggctt 90
<210> 100
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 100
ctactgtcat tcagcccaat accctaacga gaaagctgag ggtataacag gtttcaagct 60
tggcagtctg actacagagg ccactggctt 90
<210> 101
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 101
ctactgtcat tcagcccaat acccagacga gaaaagtgag ggtataacag gtttcaagct 60
tggcagtctg actacagagg ccactggctt 90
<210> 102
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 102
ctactgtcat tcagcccaat acccagacga gaaagctgag ggtataacag gtttcaagct 60
tggcagtctg actacagagg ccactggctt 90
<210> 103
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 103
ctactgtcat tcagcccaat acccagacga gaaagctgag ggtataacag gtttgtagct 60
tggcagtctg actacagagg ccactggctt 90
<210> 104
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 104
ctactgtcat tcagcccaat acccagacga gaaagctgag ggtataacag gtttcaagct 60
tggctctctg actacagagg ccactggctt 90
<210> 105
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 105
ctactgtcat tcagcccaat acccagacga gaaagctgag ggtataacag gtttcaagct 60
tggcagtctg actagtgagg ccactggctt 90
<210> 106
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 106
cactttatat ttccctgctt aaacagtccc ccgagggtgg gtgcggaaaa ggctctacac 60
ttgttatcat tccctctcca ccacaggcat 90
<210> 107
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 107
tttgtatttg ggttttttta aaacctccac tctacagtta agaattctaa ggcacagagc 60
ttcaataatt tggtcagagc caagtagcag 90
<210> 108
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 108
ggaggttaaa cccagcagca tgactgcagt tcttaatcaa tgccccttga attgcacata 60
tgggatgaac tagaacattt tctcgatgat 90
<210> 109
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 109
ctcgatgatt cgctgtcctt gttatgatta tgttactgag ctctactgta gcacagacat 60
atgtccctat atggggcggg ggtgggggtg 90
<210> 110
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Taregt sequence
<400> 110
ggtgtcttga tcgctgggct atttctatac tgttctggct tttcggaagc agtcatttct 60
ttctattctc caagcaccag caattagctt 90
<210> 111
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 111
gcttctagtt tgctgaaact aatctgctat agacagagac tccgacgaac caattttatt 60
aggatttgat caaataaact ctctctgaca 90
<210> 112
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 112
gaaagagtaa ctaagagttt gatgtttact gagtgcatag tatgcactag atgctggccg 60
tggatgcctc atagaatcct cccaacaact 90
<210> 113
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 113
gctagatgct ggccgtggat gcctcataga atcctcccaa caaccgatga aatgactact 60
gtcattcagc ccaataccca gacgagaaag 90
<210> 114
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 114
acaggtttca agcttggcag tctgactaca gaggccactg gctttacccc tgggttagtc 60
tgcctctgta ggattggggg cacgtaattt 90
<210> 115
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 115
ttagtctgcc tctgtaggat tgggggcacg taattttgct gtttaaggtc tcatttgcct 60
tcttagagat cacaagccaa agctttttat 90
<210> 116
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 116
ggaagcccag agggcatctt gtggctcggg agtagctctc tgctaccttc tcagctctgc 60
tgacaatact tgagattttc agatgtcacc 90
<210> 117
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 117
tcagctctgc tgacaatact tgagattttc agatgtcacc aaccagcaag agagcttgat 60
atgactgtat atagtatagt cataaagaac 90
<210> 118
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 118
cataaagaac ctgaacttga ccatatactt atgtcatgtg gaaatcttct catagcttca 60
gatagattat atctggagtg aagaatcctg 90
<210> 119
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 119
gtggaaaatt tctcatagct tcagatagat tatatctgga gtgagcaatc ctgccaccta 60
tgtatctggc atagtgtgag tcctcataaa 90
<210> 120
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 120
ggtttgaagg gcaacaaaat agtgaacaga gtgaaaatcc ccacctagat cctgggtcca 60
gaaaaagatg ggaaacctgt ttagctcacc 90
<210> 121
<211> 30
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 121
ggccactagg gacaaaattg gtgacagaaa 30
<210> 122
<211> 50
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 122
cccacagtgg ggccactagg gacaaaattg gtgacagaaa agccccatcc 50
<210> 123
<211> 70
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 123
tcccctccac cccacagtgg ggccactagg gacaaaattg gtgacagaaa agccccatcc 60
ttaggcctcc 70
<210> 124
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 124
cttttatctg tcccctccac cccacagtgg ggccactagg gacaaaattg gtgacagaaa 60
agccccatcc ttaggcctcc tccttcctag 90
<210> 125
<211> 110
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 125
gttctgggta cttttatctg tcccctccac cccacagtgg ggccactagg gacaaaattg 60
gtgacagaaa agccccatcc ttaggcctcc tccttcctag tctcctgata 110
<210> 126
<211> 30
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 126
tttctgtcac caatggtgtc cctagtggcc 30
<210> 127
<211> 50
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 127
ggatggggct tttctgtcac caatggtgtc cctagtggcc ccactgtggg 50
<210> 128
<211> 70
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 128
ggaggcctaa ggatggggct tttctgtcac caatggtgtc cctagtggcc ccactgtggg 60
gtggagggga 70
<210> 129
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 129
ctaggaagga ggaggcctaa ggatggggct tttctgtcac caatggtgtc cctagtggcc 60
ccactgtggg gtggagggga cagataaaag 90
<210> 130
<211> 110
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 130
tatcaggaga ctaggaagga ggaggcctaa ggatggggct tttctgtcac caatggtgtc 60
cctagtggcc ccactgtggg gtggagggga cagataaaag tacccagaac 110
<210> 131
<211> 70
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 131
ttctagtaac cactcaggac aggggggttc agcccaaaaa ttcacaagaa agtggggacc 60
catgggaaat 70
<210> 132
<211> 70
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 132
cagcaagtca gcagcacagc gtgtgtgact ccgagggtgc tccgctagcc cacattgccc 60
tctgggggtg 70
<210> 133
<211> 70
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 133
gtcagactgc caagcttgaa acctgtctta ccctctactt tctcgtctgg gtattgggct 60
gaatgacagt 70
<210> 134
<211> 70
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 134
cagagctgag aagacagcag agagctactc ccgaagcaca agatgccctc tgggcttccg 60
tgaccttggc 70
<210> 135
<211> 70
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 135
ctgacaatac ttgagatttt cagatgtcac caacgaccaa gagagcttga tatgactgta 60
tatagtatag 70
<210> 136
<211> 70
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 136
cagatacata ggtggcagga ttcttcactc cagacttaat ctatctgaag ctatgagaaa 60
ttttccacat 70
<210> 137
<211> 70
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 137
tatatgattg atttgcacag ctcatctggc cagataagct gagacatccg ttcccctaca 60
agaaactctc 70
<210> 138
<211> 70
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 138
atctggccag aagagctgag acatccgttc cccttgaaga aactctcccc ggtaagtaac 60
ctctcagctg 70
<210> 139
<211> 70
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 139
aggcatctca ctggagaggg tttagttctc cttaagagaa gataagattt caagagggaa 60
gctaagactc 70
<210> 140
<211> 70
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 140
ataatataat aaaaaatgtt tcgtctgcca ccactaatga atgtcatgca ttctgggtag 60
tttagtctta 70
<210> 141
<211> 70
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 141
tttataaagt cctagaatgt atttagttgc cctcgttgaa tgcaaactgt tttatacatc 60
aataggtttt 70
<210> 142
<211> 70
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 142
gctcaacctg gccatctctg acctgttttt ccttcccact gtccccttct gggctcacta 60
tgctgccgcc 70
<210> 143
<211> 70
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 143
ttttaaagca aacacagcat ggacgacagc caggctccta tcgattgtca ggaggatgat 60
gaagaagatt 70
<210> 144
<211> 70
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 144
gcttgtcatg gtcatctgct actcgggaat cctaattact ctgcttcggt gtcgaaatga 60
gaagaagagg 70
<210> 145
<211> 70
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 145
atactgcccc cgcgaggcca cattggcaaa ccagcttggg tgttcaactt ccagacttgg 60
ccatggagaa 70
<210> 146
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 146
ctgaagaatt tcccatgggt ccccactttc ttgtgaatcc ttggagtgaa cccccctgtc 60
ctgagtggtt actagaacac acctctggac 90
<210> 147
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 147
tggaagtatc ttgccgaggt cacacagcaa gtcagcagca cagccagtgt gactccgagc 60
ctgctccgct agcccacatt gccctctggg 90
<210> 148
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 148
ctactgtcat tcagcccaat acccagacga gaaagctgag ggtataacag gtttcaagct 60
tggcagtctg actacagagg ccactggctt 90
<210> 149
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 149
ggaagcccag agggcatctt gtggctcggg agtagctctc tgctaccttc tcagctctgc 60
tgacaatact tgagattttc agatgtcacc 90
<210> 150
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 150
tcagctctgc tgacaatact tgagattttc agatgtcacc aacgcccaag agagcttgat 60
atgactgtat atagtatagt cataaagaac 90
<210> 151
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 151
gtggaaaatt tctcatagct tcagatagat tatatctgga gtgagcaatc ctgccaccta 60
tgtatctggc atagtgtgag tcctcataaa 90
<210> 152
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 152
gaaacagcat ttcctacttt tatactgtct atatgattga tttggtcagc tcatctggcc 60
agaagagctg agacatccgt tcccctacaa 90
<210> 153
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 153
ttgatttgca cagctcatct ggccagaaga gctgagacat ccgtatccct acaagaaact 60
ctccccggta agtaacctct cagctgcttg 90
<210> 154
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 154
ggagagggtt tagttctcct tagcagaaga taagatttca agatgagagc taagactcat 60
ctctctgcaa atctttcttt tgagaggtaa 90
<210> 155
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 155
taatataata aaaaatgttt cgtctgccac cacagatgaa tgtcgagcat tctgggtagt 60
ttagtcttat aaccagctgt cttgcctagt 90
<210> 156
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 156
ttaaaaacct attgatgtat aaaacagttt gcattcatgg agggtgacta aatacattct 60
aggactttat aaaagatcac tttttattta 90
<210> 157
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 157
gacatctacc tgctcaacct ggccatctct gacctgtttt tcctatttac tgtccccttc 60
tgggctcact atgctgccgc ccagtgggac 90
<210> 158
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 158
tcatcctcct gacaatcgat aggtacctgg ctgtcgtcca tgctacgttt gctttaaaag 60
ccaggacggt cacctttggg gtggtgacaa 90
<210> 159
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 159
ggctggtcct gccgctgctt gtcatggtca tctgctactc gggagaccta aaaactctgc 60
ttcggtgtcg aaatgagaag aagaggcaca 90
<210> 160
<211> 90
<212> DNA
<213> Artificial
<220>
<223> Target sequence
<400> 160
ggcaagcctt gggtcatact gcccccgcga ggccacattg gcaagtcagc aagggtgttc 60
aacttccaga cttggccatg gagaagacat 90
<210> 161
<211> 59
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 161
acactctttc cctacacgac gctcttccga tctcgtgatt ttgcagtgtg cgttactcc 59
<210> 162
<211> 59
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 162
acactctttc cctacacgac gctcttccga tctacatcgt ttgcagtgtg cgttactcc 59
<210> 163
<211> 59
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 163
acactctttc cctacacgac gctcttccga tctgcctaat ttgcagtgtg cgttactcc 59
<210> 164
<211> 59
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 164
acactctttc cctacacgac gctcttccga tcttggtcat ttgcagtgtg cgttactcc 59
<210> 165
<211> 54
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 165
ctcggcattc ctgctgaacc gctcttccga tctccaagca actaagtcac agca 54
<210> 166
<211> 58
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 166
acactctttc cctacacgac gctcttccga tctcgtgata tgaggaaatg gaagcttg 58
<210> 167
<211> 58
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 167
acactctttc cctacacgac gctcttccga tctacatcga tgaggaaatg gaagcttg 58
<210> 168
<211> 58
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 168
acactctttc cctacacgac gctcttccga tctgcctaaa tgaggaaatg gaagcttg 58
<210> 169
<211> 58
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 169
acactctttc cctacacgac gctcttccga tcttggtcaa tgaggaaatg gaagcttg 58
<210> 170
<211> 51
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 170
ctcggcattc ctgctgaacc gctcttccga tctcattagg gtattggagg a 51
<210> 171
<211> 58
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 171
acactctttc cctacacgac gctcttccga tctcgtgata atcctcccaa caactcat 58
<210> 172
<211> 58
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 172
acactctttc cctacacgac gctcttccga tctacatcga atcctcccaa caactcat 58
<210> 173
<211> 58
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 173
acactctttc cctacacgac gctcttccga tctgcctaaa atcctcccaa caactcat 58
<210> 174
<211> 58
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 174
acactctttc cctacacgac gctcttccga tcttggtcaa atcctcccaa caactcat 58
<210> 175
<211> 52
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 175
ctcggcattc ctgctgaacc gctcttccga tctcccaatc ctacagaggc ag 52
<210> 176
<211> 58
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 176
acactctttc cctacacgac gctcttccga tctcgtgata agccaaagct ttttattc 58
<210> 177
<211> 58
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 177
acactctttc cctacacgac gctcttccga tctacatcga agccaaagct ttttattc 58
<210> 178
<211> 58
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 178
acactctttc cctacacgac gctcttccga tctgcctaaa agccaaagct ttttattc 58
<210> 179
<211> 58
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 179
acactctttc cctacacgac gctcttccga tcttggtcaa agccaaagct ttttattc 58
<210> 180
<211> 53
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 180
acactctttc cctacacgac gctcttccga tctaagccaa agctttttat tct 53
<210> 181
<211> 59
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 181
acactctttc cctacacgac gctcttccga tctcgtgata tcttgtggct cgggagtag 59
<210> 182
<211> 59
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 182
acactctttc cctacacgac gctcttccga tctacatcga tcttgtggct cgggagtag 59
<210> 183
<211> 53
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 183
ctcggcattc ctgctgaacc gctcttccga tcttggcagg attcttcact cca 53
<210> 184
<211> 59
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 184
acactctttc cctacacgac gctcttccga tctcgtgatc tattttgttg cccttcaaa 59
<210> 185
<211> 59
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 185
acactctttc cctacacgac gctcttccga tctacatcgc tattttgttg cccttcaaa 59
<210> 186
<211> 55
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 186
ctcggcattc ctgctgaacc gctcttccga tctaacctga acttgaccat atact 55
<210> 187
<211> 59
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 187
acactctttc cctacacgac gctcttccga tctcgtgatc agctgagagg ttacttacc 59
<210> 188
<211> 59
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 188
acactctttc cctacacgac gctcttccga tctacatcgc agctgagagg ttacttacc 59
<210> 189
<211> 53
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 189
ctcggcattc ctgctgaacc gctcttccga tctaatgatt taactccacc ctc 53
<210> 190
<211> 60
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 190
acactctttc cctacacgac gctcttccga tctcgtgata ctccaccctc cttcaaaaga 60
<210> 191
<211> 60
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 191
acactctttc cctacacgac gctcttccga tctacatcga ctccaccctc cttcaaaaga 60
<210> 192
<211> 52
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 192
ctcggcattc ctgctgaacc gctcttccga tcttggtgtt tgccaaatgt ct 52
<210> 193
<211> 59
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 193
acactctttc cctacacgac gctcttccga tctcgtgatg ggcacatatt cagaaggca 59
<210> 194
<211> 59
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 194
acactctttc cctacacgac gctcttccga tctacatcgg ggcacatatt cagaaggca 59
<210> 195
<211> 56
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 195
ctcggcattc ctgctgaacc gctcttccga tctagtgaaa gactttaaag ggagca 56
<210> 196
<211> 59
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 196
acactctttc cctacacgac gctcttccga tctcgtgatc acaattaaga gttgtcata 59
<210> 197
<211> 59
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 197
acactctttc cctacacgac gctcttccga tctacatcgc acaattaaga gttgtcata 59
<210> 198
<211> 53
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 198
ctcggcattc ctgctgaacc gctcttccga tctctcagct agagcagctg aac 53
<210> 199
<211> 51
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 199
ctcggcattc ctgctgaacc gctcttccga tctgacactt gataatccat c 51
<210> 200
<211> 60
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 200
acactctttc cctacacgac gctcttccga tctacatcgt caatgtagac atctatgtag 60
<210> 201
<211> 60
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 201
acactctttc cctacacgac gctcttccga tctcgtgatt caatgtagac atctatgtag 60
<210> 202
<211> 59
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 202
acactctttc cctacacgac gctcttccga tctcgtgata ctgcaaaagg ctgaagagc 59
<210> 203
<211> 59
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 203
acactctttc cctacacgac gctcttccga tctacatcga ctgcaaaagg ctgaagagc 59
<210> 204
<211> 59
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 204
acactctttc cctacacgac gctcttccga tctgcctaaa ctgcaaaagg ctgaagagc 59
<210> 205
<211> 59
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 205
acactctttc cctacacgac gctcttccga tcttggtcaa ctgcaaaagg ctgaagagc 59
<210> 206
<211> 57
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 206
ctcggcattc ctgctgaacc gctcttccga tctgcctata aaatagagcc ctgtcaa 57
<210> 207
<211> 60
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 207
acactctttc cctacacgac gctcttccga tctcgtgatc tctattttat aggcttcttc 60
<210> 208
<211> 60
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 208
acactctttc cctacacgac gctcttccga tctacatcgc tctattttat aggcttcttc 60
<210> 209
<211> 52
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 209
ctcggcattc ctgctgaacc gctcttccga tctagccacc acccaagtga tc 52
<210> 210
<211> 60
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 210
acactctttc cctacacgac gctcttccga tctacatcgt tccagacatt aaagatagtc 60
<210> 211
<211> 60
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 211
acactctttc cctacacgac gctcttccga tctcgtgatt tccagacatt aaagatagtc 60
<210> 212
<211> 54
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 212
ctcggcattc ctgctgaacc gctcttccga tctaatcatg atggtgaaga taag 54
<210> 213
<211> 60
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 213
acactctttc cctacacgac gctcttccga tctcgtgatc cggcagagac aaacattaaa 60
<210> 214
<211> 54
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 214
acactctttc cctacacgac gctcttccga tctccggcag agacaaacat taaa 54
<210> 215
<211> 53
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 215
ctcggcattc ctgctgaacc gctcttccga tctagctagg aagccatggc aag 53
<210> 216
<211> 47
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 216
aatgatacgg cgaccaccga gatctacact ctttccctac acgacgc 47
<210> 217
<211> 50
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 217
caagcagaag acggcatacg agatcggtct cggcattcct gctgaaccgc 50
<210> 218
<211> 58
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 218
acactctttc cctacacgac gctcttccga tctagtgcat agtatgtgct agatgctg 58
<210> 219
<211> 59
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 219
gtgactggag ttcagacgtg tgctcttccg atcttgatct ctaagaaggc aaatgagac 59
<210> 220
<211> 64
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<220>
<221> misc_feature
<222> (25)..(30)
<223> n is a, c, g, or t
<400> 220
caagcagaag acggcatacg agatnnnnnn gtgactggag ttcagacgtg tgctcttccg 60
atct 64
<210> 221
<211> 58
<212> DNA
<213> Artificial
<220>
<223> PCR primer
<400> 221
aatgatacgg cgaccaccga gatctacact ctttccctac acgacgctct tccgatct 58
<210> 222
<211> 34
<212> PRT
<213> Artificial
<220>
<223> Deduced TALE template amino acid sequence
<220>
<221> misc_feature
<222> (12)..(13)
<223> Xaa can be any naturally occurring amino acid
<400> 222
Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser Xaa Xaa Gly Gly Lys
1 5 10 15
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
20 25 30
His Gly
<210> 223
<211> 246
<212> DNA
<213> Artificial
<220>
<223> TALE dimer nucleotide sequence
<400> 223
cgattactgc ctgtcttatg tcaggcccat ggccttactc ctgagcaagt cgtagctatc 60
gccagcaaca taggtgggaa acaggccctg gaaaccgtac aacgtctcct cccagtactt 120
tgtcaagcac acgggttgac accggaacaa gtggtggcga ttgcgtccca cgatggaggc 180
aagcaggcac tggagaccgt ccaacggctt cttccggttc tttgccaggc tcatgggctc 240
acgcca 246
<210> 224
<211> 82
<212> PRT
<213> Artificial
<220>
<223> Deduced TALE dimer amino acid sequence
<400> 224
Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln
1 5 10 15
Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr
20 25 30
Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro
35 40 45
Glu Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu
50 55 60
Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu
65 70 75 80
Thr Pro
Claims (1)
- 반복부 서열 100 bp 이상이 결핍된 TALEN을 세포 내로 도입하는 단계를 포함하고, 여기서, 세포에서 TALEN은 표적 DNA를 절단하고, 세포는 비상동성 단부 연결을 거치고, 이로써 변경된 DNA가 생산되는 것인, 세포에서 표적 DNA를 변경시켜 인간을 치료하는 방법.
Applications Claiming Priority (4)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201361858866P | 2013-07-26 | 2013-07-26 | |
| US61/858,866 | 2013-07-26 | ||
| PCT/US2014/048140 WO2015013583A2 (en) | 2013-07-26 | 2014-07-25 | Genome engineering |
| KR1020167004663A KR20160035017A (ko) | 2013-07-26 | 2014-07-25 | 게놈 조작 |
Related Parent Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020167004663A Division KR20160035017A (ko) | 2013-07-26 | 2014-07-25 | 게놈 조작 |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20230096134A true KR20230096134A (ko) | 2023-06-29 |
Family
ID=52390824
Family Applications (2)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020237020511A KR20230096134A (ko) | 2013-07-26 | 2014-07-25 | 게놈 조작 |
| KR1020167004663A KR20160035017A (ko) | 2013-07-26 | 2014-07-25 | 게놈 조작 |
Family Applications After (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020167004663A KR20160035017A (ko) | 2013-07-26 | 2014-07-25 | 게놈 조작 |
Country Status (14)
| Country | Link |
|---|---|
| US (4) | US10563225B2 (ko) |
| EP (4) | EP3024964B1 (ko) |
| JP (4) | JP6739335B2 (ko) |
| KR (2) | KR20230096134A (ko) |
| CN (3) | CN105473773B (ko) |
| AU (4) | AU2014293015B2 (ko) |
| BR (1) | BR112016001721B1 (ko) |
| CA (1) | CA2918540A1 (ko) |
| ES (1) | ES2715666T3 (ko) |
| HK (1) | HK1218940A1 (ko) |
| IL (2) | IL243560B (ko) |
| RU (2) | RU2688462C2 (ko) |
| SG (3) | SG11201600217QA (ko) |
| WO (1) | WO2015013583A2 (ko) |
Families Citing this family (109)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US10323236B2 (en) | 2011-07-22 | 2019-06-18 | President And Fellows Of Harvard College | Evaluation and improvement of nuclease cleavage specificity |
| SG11201406547YA (en) | 2012-04-25 | 2014-11-27 | Regeneron Pharma | Nuclease-mediated targeting with large targeting vectors |
| IL308158A (en) | 2012-12-17 | 2023-12-01 | Harvard College | RNA-guided human genome engineering |
| EP2981617B1 (en) | 2013-04-04 | 2023-07-05 | President and Fellows of Harvard College | Therapeutic uses of genome editing with crispr/cas systems |
| DK2986729T3 (en) | 2013-04-16 | 2018-10-29 | Regeneron Pharma | TARGETED MODIFICATION OF ROOT THROUGH |
| JP2016528890A (ja) | 2013-07-09 | 2016-09-23 | プレジデント アンド フェローズ オブ ハーバード カレッジ | CRISPR/Cas系を用いるゲノム編集の治療用の使用 |
| US20150044192A1 (en) | 2013-08-09 | 2015-02-12 | President And Fellows Of Harvard College | Methods for identifying a target site of a cas9 nuclease |
| US9359599B2 (en) | 2013-08-22 | 2016-06-07 | President And Fellows Of Harvard College | Engineered transcription activator-like effector (TALE) domains and uses thereof |
| US9388430B2 (en) | 2013-09-06 | 2016-07-12 | President And Fellows Of Harvard College | Cas9-recombinase fusion proteins and uses thereof |
| US9526784B2 (en) | 2013-09-06 | 2016-12-27 | President And Fellows Of Harvard College | Delivery system for functional nucleases |
| US9340799B2 (en) | 2013-09-06 | 2016-05-17 | President And Fellows Of Harvard College | MRNA-sensing switchable gRNAs |
| DE202014010413U1 (de) | 2013-09-18 | 2015-12-08 | Kymab Limited | Zellen und Organismen |
| WO2015070083A1 (en) | 2013-11-07 | 2015-05-14 | Editas Medicine,Inc. | CRISPR-RELATED METHODS AND COMPOSITIONS WITH GOVERNING gRNAS |
| RU2685914C1 (ru) | 2013-12-11 | 2019-04-23 | Регенерон Фармасьютикалс, Инк. | Способы и композиции для направленной модификации генома |
| US9840699B2 (en) | 2013-12-12 | 2017-12-12 | President And Fellows Of Harvard College | Methods for nucleic acid editing |
| EP3690044B1 (en) | 2014-02-11 | 2024-01-10 | The Regents of the University of Colorado, a body corporate | Crispr enabled multiplexed genome engineering |
| EP3114227B1 (en) | 2014-03-05 | 2021-07-21 | Editas Medicine, Inc. | Crispr/cas-related methods and compositions for treating usher syndrome and retinitis pigmentosa |
| US11339437B2 (en) | 2014-03-10 | 2022-05-24 | Editas Medicine, Inc. | Compositions and methods for treating CEP290-associated disease |
| WO2015138510A1 (en) | 2014-03-10 | 2015-09-17 | Editas Medicine., Inc. | Crispr/cas-related methods and compositions for treating leber's congenital amaurosis 10 (lca10) |
| US11141493B2 (en) | 2014-03-10 | 2021-10-12 | Editas Medicine, Inc. | Compositions and methods for treating CEP290-associated disease |
| WO2015148863A2 (en) | 2014-03-26 | 2015-10-01 | Editas Medicine, Inc. | Crispr/cas-related methods and compositions for treating sickle cell disease |
| WO2015191693A2 (en) * | 2014-06-10 | 2015-12-17 | Massachusetts Institute Of Technology | Method for gene editing |
| EP3177718B1 (en) | 2014-07-30 | 2022-03-16 | President and Fellows of Harvard College | Cas9 proteins including ligand-dependent inteins |
| MX2017004890A (es) | 2014-10-15 | 2018-02-12 | Regeneron Pharma | Metodos y composiciones para la generación o el mantenimiento de células pluripotentes. |
| WO2016065364A1 (en) * | 2014-10-24 | 2016-04-28 | Life Technologies Corporation | Compositions and methods for enhancing homologous recombination |
| WO2016073990A2 (en) | 2014-11-07 | 2016-05-12 | Editas Medicine, Inc. | Methods for improving crispr/cas-mediated genome-editing |
| NZ731962A (en) | 2014-11-21 | 2022-07-01 | Regeneron Pharma | Methods and compositions for targeted genetic modification using paired guide rnas |
| CN107250148B (zh) | 2014-12-03 | 2021-04-16 | 安捷伦科技有限公司 | 具有化学修饰的指导rna |
| WO2016094679A1 (en) | 2014-12-10 | 2016-06-16 | Regents Of The University Of Minnesota | Genetically modified cells, tissues, and organs for treating disease |
| EP3271461A1 (en) * | 2015-03-20 | 2018-01-24 | Danmarks Tekniske Universitet | Crispr/cas9 based engineering of actinomycetal genomes |
| EP4019635A1 (en) * | 2015-03-25 | 2022-06-29 | Editas Medicine, Inc. | Crispr/cas-related methods, compositions and components |
| KR102648489B1 (ko) | 2015-04-06 | 2024-03-15 | 더 보드 어브 트러스티스 어브 더 리랜드 스탠포드 주니어 유니버시티 | Crispr/cas-매개 유전자 조절을 위한 화학적으로 변형된 가이드 rna |
| SG11201708653RA (en) | 2015-04-24 | 2017-11-29 | Editas Medicine Inc | Evaluation of cas9 molecule/guide rna molecule complexes |
| JP7030522B2 (ja) * | 2015-05-11 | 2022-03-07 | エディタス・メディシン、インコーポレイテッド | 幹細胞における遺伝子編集のための最適化crispr/cas9システムおよび方法 |
| WO2016196887A1 (en) | 2015-06-03 | 2016-12-08 | Board Of Regents Of The University Of Nebraska | Dna editing using single-stranded dna |
| CN116334142A (zh) | 2015-06-09 | 2023-06-27 | 爱迪塔斯医药公司 | 用于改善移植的crispr/cas相关方法和组合物 |
| US9790490B2 (en) | 2015-06-18 | 2017-10-17 | The Broad Institute Inc. | CRISPR enzymes and systems |
| WO2016205749A1 (en) | 2015-06-18 | 2016-12-22 | The Broad Institute Inc. | Novel crispr enzymes and systems |
| CN109536474A (zh) | 2015-06-18 | 2019-03-29 | 布罗德研究所有限公司 | 降低脱靶效应的crispr酶突变 |
| US10166255B2 (en) | 2015-07-31 | 2019-01-01 | Regents Of The University Of Minnesota | Intracellular genomic transplant and methods of therapy |
| WO2017040511A1 (en) | 2015-08-31 | 2017-03-09 | Agilent Technologies, Inc. | Compounds and methods for crispr/cas-based genome editing by homologous recombination |
| US11667911B2 (en) | 2015-09-24 | 2023-06-06 | Editas Medicine, Inc. | Use of exonucleases to improve CRISPR/CAS-mediated genome editing |
| EP3365356B1 (en) | 2015-10-23 | 2023-06-28 | President and Fellows of Harvard College | Nucleobase editors and uses thereof |
| ES2953925T3 (es) * | 2015-11-04 | 2023-11-17 | Fate Therapeutics Inc | Ingeniería genómica de células pluripotentes |
| KR101885901B1 (ko) * | 2015-11-13 | 2018-08-07 | 기초과학연구원 | 5' 말단의 인산기가 제거된 rna를 포함하는 리보핵산단백질 전달용 조성물 |
| WO2017087708A1 (en) | 2015-11-19 | 2017-05-26 | The Brigham And Women's Hospital, Inc. | Lymphocyte antigen cd5-like (cd5l)-interleukin 12b (p40) heterodimers in immunity |
| EP3383409A4 (en) * | 2015-12-02 | 2019-10-02 | The Regents of The University of California | COMPOSITIONS AND METHODS FOR MODIFYING TARGET NUCLEIC ACID |
| US11441146B2 (en) * | 2016-01-11 | 2022-09-13 | Christiana Care Health Services, Inc. | Compositions and methods for improving homogeneity of DNA generated using a CRISPR/Cas9 cleavage system |
| CN106957856B (zh) * | 2016-01-12 | 2020-07-28 | 中国科学院广州生物医药与健康研究院 | 无毛模型猪的重构卵及其构建方法和模型猪的构建方法 |
| WO2017165862A1 (en) | 2016-03-25 | 2017-09-28 | Editas Medicine, Inc. | Systems and methods for treating alpha 1-antitrypsin (a1at) deficiency |
| US11597924B2 (en) | 2016-03-25 | 2023-03-07 | Editas Medicine, Inc. | Genome editing systems comprising repair-modulating enzyme molecules and methods of their use |
| WO2017180694A1 (en) | 2016-04-13 | 2017-10-19 | Editas Medicine, Inc. | Cas9 fusion molecules gene editing systems, and methods of use thereof |
| CA3026112A1 (en) | 2016-04-19 | 2017-10-26 | The Broad Institute, Inc. | Cpf1 complexes with reduced indel activity |
| WO2017184768A1 (en) | 2016-04-19 | 2017-10-26 | The Broad Institute Inc. | Novel crispr enzymes and systems |
| KR20230038804A (ko) * | 2016-04-29 | 2023-03-21 | 바스프 플랜트 사이언스 컴퍼니 게엠베하 | 표적 핵산의 변형을 위한 개선된 방법 |
| WO2017207589A1 (en) * | 2016-06-01 | 2017-12-07 | Kws Saat Se | Hybrid nucleic acid sequences for genome engineering |
| US10767175B2 (en) | 2016-06-08 | 2020-09-08 | Agilent Technologies, Inc. | High specificity genome editing using chemically modified guide RNAs |
| WO2017223538A1 (en) | 2016-06-24 | 2017-12-28 | The Regents Of The University Of Colorado, A Body Corporate | Methods for generating barcoded combinatorial libraries |
| US11471462B2 (en) | 2016-06-27 | 2022-10-18 | The Broad Institute, Inc. | Compositions and methods for detecting and treating diabetes |
| US11359234B2 (en) | 2016-07-01 | 2022-06-14 | Microsoft Technology Licensing, Llc | Barcoding sequences for identification of gene expression |
| US20180004537A1 (en) * | 2016-07-01 | 2018-01-04 | Microsoft Technology Licensing, Llc | Molecular State Machines |
| US11566263B2 (en) | 2016-08-02 | 2023-01-31 | Editas Medicine, Inc. | Compositions and methods for treating CEP290 associated disease |
| GB2568182A (en) | 2016-08-03 | 2019-05-08 | Harvard College | Adenosine nucleobase editors and uses thereof |
| AU2017308889B2 (en) | 2016-08-09 | 2023-11-09 | President And Fellows Of Harvard College | Programmable Cas9-recombinase fusion proteins and uses thereof |
| US11542509B2 (en) | 2016-08-24 | 2023-01-03 | President And Fellows Of Harvard College | Incorporation of unnatural amino acids into proteins using base editing |
| EP3510151A4 (en) | 2016-09-09 | 2020-04-15 | The Board of Trustees of the Leland Stanford Junior University | HIGH-THROUGHPUT PRECISION GENE EDITING |
| KR102622411B1 (ko) | 2016-10-14 | 2024-01-10 | 프레지던트 앤드 펠로우즈 오브 하바드 칼리지 | 핵염기 에디터의 aav 전달 |
| CN110520530A (zh) | 2016-10-18 | 2019-11-29 | 明尼苏达大学董事会 | 肿瘤浸润性淋巴细胞和治疗方法 |
| KR20190089175A (ko) * | 2016-11-18 | 2019-07-30 | 진에딧 인코포레이티드 | 표적 핵산 변형을 위한 조성물 및 방법 |
| WO2018119359A1 (en) | 2016-12-23 | 2018-06-28 | President And Fellows Of Harvard College | Editing of ccr5 receptor gene to protect against hiv infection |
| US11530388B2 (en) * | 2017-02-14 | 2022-12-20 | University of Pittsburgh—of the Commonwealth System of Higher Education | Methods of engineering human induced pluripotent stem cells to produce liver tissue |
| US11898179B2 (en) | 2017-03-09 | 2024-02-13 | President And Fellows Of Harvard College | Suppression of pain by gene editing |
| WO2018165629A1 (en) | 2017-03-10 | 2018-09-13 | President And Fellows Of Harvard College | Cytosine to guanine base editor |
| EP3596217A1 (en) | 2017-03-14 | 2020-01-22 | Editas Medicine, Inc. | Systems and methods for the treatment of hemoglobinopathies |
| EP3601562A1 (en) | 2017-03-23 | 2020-02-05 | President and Fellows of Harvard College | Nucleobase editors comprising nucleic acid programmable dna binding proteins |
| US20210115407A1 (en) | 2017-04-12 | 2021-04-22 | The Broad Institute, Inc. | Respiratory and sweat gland ionocytes |
| WO2018195486A1 (en) | 2017-04-21 | 2018-10-25 | The Broad Institute, Inc. | Targeted delivery to beta cells |
| EP3615672A1 (en) | 2017-04-28 | 2020-03-04 | Editas Medicine, Inc. | Methods and systems for analyzing guide rna molecules |
| WO2018204777A2 (en) | 2017-05-05 | 2018-11-08 | The Broad Institute, Inc. | Methods for identification and modification of lncrna associated with target genotypes and phenotypes |
| EP3622070A2 (en) | 2017-05-10 | 2020-03-18 | Editas Medicine, Inc. | Crispr/rna-guided nuclease systems and methods |
| WO2018209320A1 (en) | 2017-05-12 | 2018-11-15 | President And Fellows Of Harvard College | Aptazyme-embedded guide rnas for use with crispr-cas9 in genome editing and transcriptional activation |
| AU2018279829B2 (en) | 2017-06-09 | 2024-01-04 | Editas Medicine, Inc. | Engineered Cas9 nucleases |
| US9982279B1 (en) | 2017-06-23 | 2018-05-29 | Inscripta, Inc. | Nucleic acid-guided nucleases |
| US10011849B1 (en) | 2017-06-23 | 2018-07-03 | Inscripta, Inc. | Nucleic acid-guided nucleases |
| WO2019006418A2 (en) | 2017-06-30 | 2019-01-03 | Intima Bioscience, Inc. | ADENO-ASSOCIATED VIRAL VECTORS FOR GENE THERAPY |
| WO2019014564A1 (en) | 2017-07-14 | 2019-01-17 | Editas Medicine, Inc. | SYSTEMS AND METHODS OF TARGETED INTEGRATION AND GENOME EDITING AND DETECTION THEREOF WITH INTEGRATED PRIMING SITES |
| US11732274B2 (en) | 2017-07-28 | 2023-08-22 | President And Fellows Of Harvard College | Methods and compositions for evolving base editors using phage-assisted continuous evolution (PACE) |
| MX2020001178A (es) | 2017-07-31 | 2020-09-25 | Regeneron Pharma | Celulas madre embrionarias de raton transgenico con cas y ratones y usos de los mismos. |
| EP3676376A2 (en) | 2017-08-30 | 2020-07-08 | President and Fellows of Harvard College | High efficiency base editors comprising gam |
| CN111344403A (zh) * | 2017-09-15 | 2020-06-26 | 利兰斯坦福初级大学董事会 | 遗传工程细胞的多元性产生和条形码编制 |
| KR20200121782A (ko) | 2017-10-16 | 2020-10-26 | 더 브로드 인스티튜트, 인코퍼레이티드 | 아데노신 염기 편집제의 용도 |
| US11268092B2 (en) | 2018-01-12 | 2022-03-08 | GenEdit, Inc. | Structure-engineered guide RNA |
| CN112272516B (zh) | 2018-04-06 | 2023-05-30 | 儿童医疗中心有限公司 | 用于体细胞重新编程和调整印记的组合物和方法 |
| GB2589246A (en) | 2018-05-16 | 2021-05-26 | Synthego Corp | Methods and systems for guide RNA design and use |
| US20210246505A1 (en) * | 2018-06-21 | 2021-08-12 | Memorial Sloan Kettering Cancer Center | Compositions & methods for monitoring dna repair |
| KR20210053898A (ko) | 2018-07-31 | 2021-05-12 | 더 브로드 인스티튜트, 인코퍼레이티드 | 신규 crispr 효소 및 시스템 |
| CA3121268A1 (en) | 2018-12-10 | 2020-06-18 | Amgen Inc. | Mutated piggybac transposase |
| BR112021018606A2 (pt) | 2019-03-19 | 2021-11-23 | Harvard College | Métodos e composições para editar sequências de nucleotídeos |
| CN115175996A (zh) | 2019-09-20 | 2022-10-11 | 博德研究所 | 新颖vi型crispr酶和系统 |
| EP4146797A1 (en) | 2020-05-06 | 2023-03-15 | Orchard Therapeutics (Europe) Limited | Treatment for neurodegenerative diseases |
| DE112021002672T5 (de) | 2020-05-08 | 2023-04-13 | President And Fellows Of Harvard College | Vefahren und zusammensetzungen zum gleichzeitigen editieren beider stränge einer doppelsträngigen nukleotid-zielsequenz |
| EP4232583A1 (en) | 2020-10-21 | 2023-08-30 | Massachusetts Institute of Technology | Systems, methods, and compositions for site-specific genetic engineering using programmable addition via site-specific targeting elements (paste) |
| TW202233660A (zh) | 2020-10-30 | 2022-09-01 | 美商安進公司 | 過表現胰島素樣生長因子受體突變體以調節igf補充 |
| KR20240055811A (ko) | 2021-09-10 | 2024-04-29 | 애질런트 테크놀로지스, 인크. | 프라임 편집을 위한 화학적 변형을 갖는 가이드 rna |
| CA3237300A1 (en) | 2021-11-01 | 2023-05-04 | Tome Biosciences, Inc. | Single construct platform for simultaneous delivery of gene editing machinery and nucleic acid cargo |
| WO2023122764A1 (en) | 2021-12-22 | 2023-06-29 | Tome Biosciences, Inc. | Co-delivery of a gene editor construct and a donor template |
| WO2023205744A1 (en) | 2022-04-20 | 2023-10-26 | Tome Biosciences, Inc. | Programmable gene insertion compositions |
| WO2023225670A2 (en) | 2022-05-20 | 2023-11-23 | Tome Biosciences, Inc. | Ex vivo programmable gene insertion |
| WO2024020587A2 (en) | 2022-07-22 | 2024-01-25 | Tome Biosciences, Inc. | Pleiopluripotent stem cell programmable gene insertion |
Family Cites Families (43)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US5641670A (en) * | 1991-11-05 | 1997-06-24 | Transkaryotic Therapies, Inc. | Protein production and protein delivery |
| US6689558B2 (en) | 2000-02-08 | 2004-02-10 | Sangamo Biosciences, Inc. | Cells for drug discovery |
| EP1280928B1 (en) * | 2000-05-10 | 2016-11-30 | Board Of Supervisors Of Louisiana State University And Agricultural And Mechanical College | Resistance to acetohydroxyacid synthase-inhibiting herbicides |
| CA2474486C (en) | 2002-01-23 | 2013-05-14 | The University Of Utah Research Foundation | Targeted chromosomal mutagenesis using zinc finger nucleases |
| KR100766952B1 (ko) | 2002-12-09 | 2007-10-17 | 주식회사 툴젠 | 조절성 징크 핑거 단백질 |
| JP2007521823A (ja) | 2004-02-13 | 2007-08-09 | ノボザイムス アクティーゼルスカブ | プロテアーゼ変異型 |
| DE102005062662A1 (de) | 2005-12-23 | 2007-06-28 | Basf Ag | Verfahren zur Herstellung optisch aktiver Alkohole |
| BRPI0808704B1 (pt) | 2007-03-02 | 2022-01-18 | Dupont Nutrition Biosciences Aps | Método para gerar uma cultura inicial compreendendo pelo menos duas cepas variantes resistentes a bacteriófagos, cultura iniciadora e método de fermentação |
| US8546553B2 (en) | 2008-07-25 | 2013-10-01 | University Of Georgia Research Foundation, Inc. | Prokaryotic RNAi-like system and methods of use |
| US20100076057A1 (en) | 2008-09-23 | 2010-03-25 | Northwestern University | TARGET DNA INTERFERENCE WITH crRNA |
| US9404098B2 (en) | 2008-11-06 | 2016-08-02 | University Of Georgia Research Foundation, Inc. | Method for cleaving a target RNA using a Cas6 polypeptide |
| PH12012500097A1 (en) * | 2009-07-21 | 2011-01-27 | Shanghai Inst Organic Chem | Potent small molecule inhibitors of autophagy, and methods of use thereof |
| PT2816112T (pt) | 2009-12-10 | 2018-11-20 | Univ Iowa State Res Found Inc | Modificação do adn modificada pelo efector tal |
| NO332016B1 (no) | 2009-12-29 | 2012-05-21 | Stiftelsen Sintef | Prøvebehandlingskassett og fremgangsmåte for å behandle og/eller analysere en prøve under sentrifugalkraft |
| GB201000591D0 (en) | 2010-01-14 | 2010-03-03 | Ucb Pharma Sa | Bacterial hoist strain |
| MX336731B (es) * | 2010-01-28 | 2016-01-28 | Harvard College | Composiciones y metodos para potenciar la actividad de proteasoma. |
| US10087431B2 (en) | 2010-03-10 | 2018-10-02 | The Regents Of The University Of California | Methods of generating nucleic acid fragments |
| BR112012028805A2 (pt) | 2010-05-10 | 2019-09-24 | The Regents Of The Univ Of California E Nereus Pharmaceuticals Inc | composições de endorribonuclease e métodos de uso das mesmas. |
| CN103025344B (zh) | 2010-05-17 | 2016-06-29 | 桑格摩生物科学股份有限公司 | 新型dna-结合蛋白及其用途 |
| US20140113376A1 (en) | 2011-06-01 | 2014-04-24 | Rotem Sorek | Compositions and methods for downregulating prokaryotic genes |
| CA2841541C (en) | 2011-07-25 | 2019-11-12 | Sangamo Biosciences, Inc. | Methods and compositions for alteration of a cystic fibrosis transmembrane conductance regulator (cftr) gene |
| US8450107B1 (en) | 2011-11-30 | 2013-05-28 | The Broad Institute Inc. | Nucleotide-specific recognition sequences for designer TAL effectors |
| GB201122458D0 (en) | 2011-12-30 | 2012-02-08 | Univ Wageningen | Modified cascade ribonucleoproteins and uses thereof |
| RU2650811C2 (ru) | 2012-02-24 | 2018-04-17 | Фред Хатчинсон Кэнсер Рисерч Сентер | Композиции и способы лечения гемоглобинопатии |
| EP2820159B1 (en) | 2012-02-29 | 2019-10-23 | Sangamo Therapeutics, Inc. | Methods and compositions for treating huntington's disease |
| WO2013141680A1 (en) | 2012-03-20 | 2013-09-26 | Vilnius University | RNA-DIRECTED DNA CLEAVAGE BY THE Cas9-crRNA COMPLEX |
| US9637739B2 (en) | 2012-03-20 | 2017-05-02 | Vilnius University | RNA-directed DNA cleavage by the Cas9-crRNA complex |
| ES2960803T3 (es) | 2012-05-25 | 2024-03-06 | Univ California | Métodos y composiciones para la modificación de ADN diana dirigida por RNA y para la modulación de la transcripción dirigida por RNA |
| EP2880171B1 (en) | 2012-08-03 | 2018-10-03 | The Regents of The University of California | Methods and compositions for controlling gene expression by rna processing |
| ES2926021T3 (es) | 2012-10-23 | 2022-10-21 | Toolgen Inc | Composición para escindir un ADN objetivo que comprende un ARN guía específico para el ADN objetivo y ácido nucleico codificador de proteína Cas o proteína Cas, y uso de la misma |
| EP3617309A3 (en) | 2012-12-06 | 2020-05-06 | Sigma Aldrich Co. LLC | Crispr-based genome modification and regulation |
| SG10201912328UA (en) | 2012-12-12 | 2020-02-27 | Broad Inst Inc | Delivery, Engineering and Optimization of Systems, Methods and Compositions for Sequence Manipulation and Therapeutic Applications |
| CN113355357A (zh) | 2012-12-12 | 2021-09-07 | 布罗德研究所有限公司 | 对用于序列操纵的改进的系统、方法和酶组合物进行的工程化和优化 |
| US8697359B1 (en) | 2012-12-12 | 2014-04-15 | The Broad Institute, Inc. | CRISPR-Cas systems and methods for altering expression of gene products |
| AU2014235794A1 (en) | 2013-03-14 | 2015-10-22 | Caribou Biosciences, Inc. | Compositions and methods of nucleic acid-targeting nucleic acids |
| US9234213B2 (en) | 2013-03-15 | 2016-01-12 | System Biosciences, Llc | Compositions and methods directed to CRISPR/Cas genomic engineering systems |
| US20140349400A1 (en) | 2013-03-15 | 2014-11-27 | Massachusetts Institute Of Technology | Programmable Modification of DNA |
| EP2981617B1 (en) | 2013-04-04 | 2023-07-05 | President and Fellows of Harvard College | Therapeutic uses of genome editing with crispr/cas systems |
| US20140349405A1 (en) | 2013-05-22 | 2014-11-27 | Wisconsin Alumni Research Foundation | Rna-directed dna cleavage and gene editing by cas9 enzyme from neisseria meningitidis |
| US11685935B2 (en) | 2013-05-29 | 2023-06-27 | Cellectis | Compact scaffold of Cas9 in the type II CRISPR system |
| WO2014198911A1 (en) | 2013-06-14 | 2014-12-18 | Cellectis | Improved polynucleotide sequences encoding tale repeats |
| WO2014204725A1 (en) | 2013-06-17 | 2014-12-24 | The Broad Institute Inc. | Optimized crispr-cas double nickase systems, methods and compositions for sequence manipulation |
| CA2933134A1 (en) | 2013-12-13 | 2015-06-18 | Cellectis | Cas9 nuclease platform for microalgae genome engineering |
-
2014
- 2014-06-30 US US14/319,380 patent/US10563225B2/en active Active
- 2014-06-30 US US14/319,498 patent/US11306328B2/en active Active
- 2014-07-25 SG SG11201600217QA patent/SG11201600217QA/en unknown
- 2014-07-25 SG SG10201800259WA patent/SG10201800259WA/en unknown
- 2014-07-25 EP EP14829377.2A patent/EP3024964B1/en active Active
- 2014-07-25 CN CN201480042306.1A patent/CN105473773B/zh active Active
- 2014-07-25 EP EP21176056.6A patent/EP3916099B1/en active Active
- 2014-07-25 WO PCT/US2014/048140 patent/WO2015013583A2/en active Application Filing
- 2014-07-25 AU AU2014293015A patent/AU2014293015B2/en active Active
- 2014-07-25 KR KR1020237020511A patent/KR20230096134A/ko not_active Application Discontinuation
- 2014-07-25 RU RU2016106649A patent/RU2688462C2/ru active
- 2014-07-25 CN CN202010094041.7A patent/CN111304230A/zh active Pending
- 2014-07-25 KR KR1020167004663A patent/KR20160035017A/ko not_active IP Right Cessation
- 2014-07-25 EP EP18212761.3A patent/EP3483277B1/en active Active
- 2014-07-25 EP EP23164284.4A patent/EP4234703A3/en active Pending
- 2014-07-25 RU RU2019114217A patent/RU2764757C2/ru active
- 2014-07-25 SG SG10201913000PA patent/SG10201913000PA/en unknown
- 2014-07-25 ES ES14829377T patent/ES2715666T3/es active Active
- 2014-07-25 US US14/907,605 patent/US9914939B2/en active Active
- 2014-07-25 JP JP2016530062A patent/JP6739335B2/ja active Active
- 2014-07-25 CA CA2918540A patent/CA2918540A1/en active Pending
- 2014-07-25 BR BR112016001721-8A patent/BR112016001721B1/pt active IP Right Grant
- 2014-07-25 CN CN202410183863.0A patent/CN118028379A/zh active Pending
-
2016
- 2016-01-11 IL IL243560A patent/IL243560B/en active IP Right Grant
- 2016-06-14 HK HK16106842.9A patent/HK1218940A1/zh unknown
-
2018
- 2018-12-12 AU AU2018278911A patent/AU2018278911C1/en active Active
-
2020
- 2020-01-22 IL IL272193A patent/IL272193B/en unknown
- 2020-02-21 JP JP2020028641A patent/JP6993063B2/ja active Active
-
2021
- 2021-04-27 AU AU2021202581A patent/AU2021202581B2/en active Active
- 2021-12-07 JP JP2021198648A patent/JP7419328B2/ja active Active
-
2022
- 2022-04-07 US US17/715,177 patent/US20220235382A1/en active Pending
-
2023
- 2023-10-13 AU AU2023248167A patent/AU2023248167A1/en active Pending
-
2024
- 2024-01-10 JP JP2024002064A patent/JP2024029218A/ja active Pending
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP7419328B2 (ja) | ゲノムエンジニアリング | |
| KR20220100090A (ko) | 대형 유전자 절제 및 삽입 | |
| RU2812848C2 (ru) | Геномная инженерия | |
| NZ754902A (en) | Genome engineering | |
| NZ754904B2 (en) | Genome engineering | |
| NZ754903B2 (en) | Genome engineering | |
| NZ754902B2 (en) | Genome engineering | |
| NZ716606B2 (en) | Genome engineering |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A107 | Divisional application of patent | ||
| E902 | Notification of reason for refusal |