KR20170041907A - 매니폴드 및 초평면을 이용한 생물학적 데이터의 컴퓨터 분석법 - Google Patents
매니폴드 및 초평면을 이용한 생물학적 데이터의 컴퓨터 분석법 Download PDFInfo
- Publication number
- KR20170041907A KR20170041907A KR1020177007002A KR20177007002A KR20170041907A KR 20170041907 A KR20170041907 A KR 20170041907A KR 1020177007002 A KR1020177007002 A KR 1020177007002A KR 20177007002 A KR20177007002 A KR 20177007002A KR 20170041907 A KR20170041907 A KR 20170041907A
- Authority
- KR
- South Korea
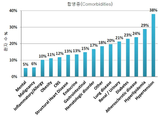
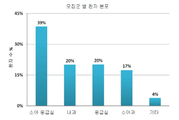
- Prior art keywords
- subject
- bacterial
- infection
- viral
- trail
- Prior art date
Links
Images
Classifications
-
- G06F19/10—
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B99/00—Subject matter not provided for in other groups of this subclass
-
- G—PHYSICS
- G01—MEASURING; TESTING
- G01N—INVESTIGATING OR ANALYSING MATERIALS BY DETERMINING THEIR CHEMICAL OR PHYSICAL PROPERTIES
- G01N33/00—Investigating or analysing materials by specific methods not covered by groups G01N1/00 - G01N31/00
- G01N33/48—Biological material, e.g. blood, urine; Haemocytometers
- G01N33/50—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing
- G01N33/53—Immunoassay; Biospecific binding assay; Materials therefor
- G01N33/569—Immunoassay; Biospecific binding assay; Materials therefor for microorganisms, e.g. protozoa, bacteria, viruses
-
- G—PHYSICS
- G06—COMPUTING; CALCULATING OR COUNTING
- G06F—ELECTRIC DIGITAL DATA PROCESSING
- G06F17/00—Digital computing or data processing equipment or methods, specially adapted for specific functions
- G06F17/10—Complex mathematical operations
- G06F17/18—Complex mathematical operations for evaluating statistical data, e.g. average values, frequency distributions, probability functions, regression analysis
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B20/00—ICT specially adapted for functional genomics or proteomics, e.g. genotype-phenotype associations
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B20/00—ICT specially adapted for functional genomics or proteomics, e.g. genotype-phenotype associations
- G16B20/20—Allele or variant detection, e.g. single nucleotide polymorphism [SNP] detection
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B25/00—ICT specially adapted for hybridisation; ICT specially adapted for gene or protein expression
- G16B25/10—Gene or protein expression profiling; Expression-ratio estimation or normalisation
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B40/00—ICT specially adapted for biostatistics; ICT specially adapted for bioinformatics-related machine learning or data mining, e.g. knowledge discovery or pattern finding
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B40/00—ICT specially adapted for biostatistics; ICT specially adapted for bioinformatics-related machine learning or data mining, e.g. knowledge discovery or pattern finding
- G16B40/20—Supervised data analysis
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B40/00—ICT specially adapted for biostatistics; ICT specially adapted for bioinformatics-related machine learning or data mining, e.g. knowledge discovery or pattern finding
- G16B40/30—Unsupervised data analysis
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B5/00—ICT specially adapted for modelling or simulations in systems biology, e.g. gene-regulatory networks, protein interaction networks or metabolic networks
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B5/00—ICT specially adapted for modelling or simulations in systems biology, e.g. gene-regulatory networks, protein interaction networks or metabolic networks
- G16B5/20—Probabilistic models
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16H—HEALTHCARE INFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR THE HANDLING OR PROCESSING OF MEDICAL OR HEALTHCARE DATA
- G16H50/00—ICT specially adapted for medical diagnosis, medical simulation or medical data mining; ICT specially adapted for detecting, monitoring or modelling epidemics or pandemics
- G16H50/50—ICT specially adapted for medical diagnosis, medical simulation or medical data mining; ICT specially adapted for detecting, monitoring or modelling epidemics or pandemics for simulation or modelling of medical disorders
-
- G—PHYSICS
- G01—MEASURING; TESTING
- G01N—INVESTIGATING OR ANALYSING MATERIALS BY DETERMINING THEIR CHEMICAL OR PHYSICAL PROPERTIES
- G01N33/00—Investigating or analysing materials by specific methods not covered by groups G01N1/00 - G01N31/00
- G01N33/48—Biological material, e.g. blood, urine; Haemocytometers
- G01N33/50—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing
- G01N33/53—Immunoassay; Biospecific binding assay; Materials therefor
- G01N33/569—Immunoassay; Biospecific binding assay; Materials therefor for microorganisms, e.g. protozoa, bacteria, viruses
- G01N33/56911—Bacteria
-
- G—PHYSICS
- G01—MEASURING; TESTING
- G01N—INVESTIGATING OR ANALYSING MATERIALS BY DETERMINING THEIR CHEMICAL OR PHYSICAL PROPERTIES
- G01N33/00—Investigating or analysing materials by specific methods not covered by groups G01N1/00 - G01N31/00
- G01N33/48—Biological material, e.g. blood, urine; Haemocytometers
- G01N33/50—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing
- G01N33/53—Immunoassay; Biospecific binding assay; Materials therefor
- G01N33/569—Immunoassay; Biospecific binding assay; Materials therefor for microorganisms, e.g. protozoa, bacteria, viruses
- G01N33/56983—Viruses
-
- G—PHYSICS
- G16—INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR SPECIFIC APPLICATION FIELDS
- G16B—BIOINFORMATICS, i.e. INFORMATION AND COMMUNICATION TECHNOLOGY [ICT] SPECIALLY ADAPTED FOR GENETIC OR PROTEIN-RELATED DATA PROCESSING IN COMPUTATIONAL MOLECULAR BIOLOGY
- G16B25/00—ICT specially adapted for hybridisation; ICT specially adapted for gene or protein expression
-
- Y—GENERAL TAGGING OF NEW TECHNOLOGICAL DEVELOPMENTS; GENERAL TAGGING OF CROSS-SECTIONAL TECHNOLOGIES SPANNING OVER SEVERAL SECTIONS OF THE IPC; TECHNICAL SUBJECTS COVERED BY FORMER USPC CROSS-REFERENCE ART COLLECTIONS [XRACs] AND DIGESTS
- Y02—TECHNOLOGIES OR APPLICATIONS FOR MITIGATION OR ADAPTATION AGAINST CLIMATE CHANGE
- Y02A—TECHNOLOGIES FOR ADAPTATION TO CLIMATE CHANGE
- Y02A90/00—Technologies having an indirect contribution to adaptation to climate change
- Y02A90/10—Information and communication technologies [ICT] supporting adaptation to climate change, e.g. for weather forecasting or climate simulation
Landscapes
- Health & Medical Sciences (AREA)
- Engineering & Computer Science (AREA)
- Life Sciences & Earth Sciences (AREA)
- Physics & Mathematics (AREA)
- Medical Informatics (AREA)
- Bioinformatics & Cheminformatics (AREA)
- General Health & Medical Sciences (AREA)
- Biotechnology (AREA)
- Theoretical Computer Science (AREA)
- Bioinformatics & Computational Biology (AREA)
- Evolutionary Biology (AREA)
- Spectroscopy & Molecular Physics (AREA)
- Biophysics (AREA)
- Data Mining & Analysis (AREA)
- Molecular Biology (AREA)
- Public Health (AREA)
- Genetics & Genomics (AREA)
- Databases & Information Systems (AREA)
- Chemical & Material Sciences (AREA)
- Epidemiology (AREA)
- Software Systems (AREA)
- Immunology (AREA)
- Computer Vision & Pattern Recognition (AREA)
- Bioethics (AREA)
- Evolutionary Computation (AREA)
- Artificial Intelligence (AREA)
- Analytical Chemistry (AREA)
- Biomedical Technology (AREA)
- General Physics & Mathematics (AREA)
- Urology & Nephrology (AREA)
- Hematology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Physiology (AREA)
- Pathology (AREA)
- Probability & Statistics with Applications (AREA)
- Virology (AREA)
- Microbiology (AREA)
- Tropical Medicine & Parasitology (AREA)
- Biochemistry (AREA)
- Medicinal Chemistry (AREA)
Abstract
대상체의 혈액 내 복수의 폴리펩타이드의 발현값을 포함하는 생물학적 데이터를 분석하는 방법. 상기 방법은 곡선의 한 세그먼트 및 한 방향으로 정의되는 한 축 간의 거리를 계산하는 단계를 포함하고, 상기 거리는 방향에 따른 좌표에 의해 정의되는 곡선 상의 한 점에서 계산된다. 상기 방법은 상기 거리를 대상체의 박테리아성 감염 유무 또는 가능성과 연관시키는 단계를 더 포함한다. 상기 좌표는 발현값들의 조합에 의해 정의되고, 상기 세그먼트의 90% 이상은 하한선 및 상한선 사이에 있다.
Description
본 발명은 컴퓨터 분석법에 관한 것이고, 더 구체적으로, 예를 들어 박테리아성 감염 및 비-박테리아성 감염 간, 및/또는 박테리아성 감염 및 바이러스성 감염 간, 및/또는 감염성 및 비감염성 질병 간의 차이를 구별하는 목적의 생물학적 데이터의 컴퓨터 분석법에 관한 것이다.
항생제 (Abx)는 세계에서 가장 많이 처방되는 약 250-300억 달러 규모의 약물이다. 항생제는 또한 모든 약물 중 상당 부분(40-70%)이 잘못 처방되어 세계에서 가장 오용되는 약물이다(Linder, J.A. and R.S. Stafford 2001; Scott, J. G. and D. Cohen, et al. 2001; Davey, P. and E. Brown, et al. 2006; Cadieux, G. and R. Tamblyn, et al. 2007; Pulcini, C. and E. Cua, et al. 2007), ("CDC - Get Smart: Fast Facts About Antibiotic Resistance" 2011).
항생제 오용의 한 유형은 항생제가 효과적이지 않은 바이러스성 감염과 같은 비 박테리아성 질병에 약물을 투여하는 것이다. 예를 들어, 미국 질병 관리 센터 (CDC)에 따르면 미국에서 독감을 치료하기 위해 매년 6천만건 이상의 잘못된 항생제 처방이 이루어진다. 항생제 처방 남용의 건강 관리 및 경제적 결과를 다음과 같다: (i) 연간 100억 달러 이상으로 추정되는, 세계적으로 불필요하게 처방된 항생제 비용, (ii) 불필요한 항생제 처방으로 인한 부작용으로, 합병증 및 입원 기간 연장으로 인한 의료의 질 감소 (예, 알레르기 반응, 항생제 관련 설사 및 장내 효모 등), (iii) 항생제 남용으로 인한 내성 균주의 출현 (CDC는 박테리아성의 항생제 내성이 “21 세기의 세계에서 가장 시급한 건강 문제 중 하나라고 주장했다(Arias, C.A. and B.E. Murray 2009; "CDC - About Antimicrobial Resistance" 2011)).
항생제 처방 역시 일반적이지 않다. 예를 들어, 미국에서 입원한 성인 박테리아성 폐렴 환자의 15%까지 조기 치료로 생명을 구하고 합병증을 줄일 수 있음에도 불구하고 항생제 치료가 지연되거나 전혀 받지 못한다.
감염성 질병 진단 기술은 항생제 오용과 관련된 건강 및 경제적 부담을 감소시켜준다. 이상적으로, 이런 기술은 (i) 박테리아성 및 바이러스성 감염을 정확히 구별하고; (ii) 신속하고(수 분내); (iii) 신체의 자연적 식물군의 일부인 병원성 및 비-병원성 박테리아를 구별할 수 있고; (iv) 혼합된 공동 감염 및 순수 바이러스성 감염을 구별하고, 그리고 (v) 병원체가 접근 불가능한 경우에 적용 가능하여야 한다(예, 부비동염, 폐렴, 중이염, 기관지염 등).
현재 해결책(예, 배양, PCR 및 면역분석법)은 다음 요구 사항을 모두 충족시키지 못한다: (i) 몇몇 분석법은 진단 정확성이 낮고(예, 낮은 민감도 또는 특이성)(Uyeki et al. 2009), 박테리아성 또는 바이러스성 균주로 한정된다; (ii) 상기 분석법들은 종종 수 시간에서 수일이 소요된다; (iii) 병원성 및 비 병원성 박테리아를 구별하지 못해(Del Mar, C 1992), 거짓 양성(false positives)을 초래한다; (iv) 혼합된 바이러스성 감염 및 순수 바이러스성 감염을 구별하지 못하고 (v) 병을 야기하는 병원균의 흔적이 있는 감염 부위를 직접 표본 추출해야 하므로 종종 접근하기 어려운 조직에 있는 병원체의 경우에 진단이 금지된다.
결국, 여전히 진단적 의견차이가 존재하기 때문에, 의사는 항생제를 과용(the "Just-in-case-approach")하거나 항생제를 처방하지 않도록(the "Wait-and-see-approach") 유도하고(Little, P.S. and I. Williamson 1994; Little, P. 2005; Spiro, D. M. and K. Y. Tay, et al. 2006), 이는 두 경우 모두 건강 및 경제적 결과를 가져온다.
따라서, 이러한 문제를 해결할 수 있는 박테리아성(혼합된 박테리아성 및 바이러스성 감염 포함), 바이러스성 및 비-박테리아성, 비-바이러스성 질병 환자를 정확하게 구별할 수 있는 신속한 방법이 필요하다.
WO 2013/117746은 박테리아성 및 바이러스성 감염을 구별하기 위한 시그니쳐 및 결정인자를 개시한다.
본 발명의 일부 실시예에 따르면, 대상체의 혈액 중의 복수의 폴리펩타이드의 발현값을 포함하는 생물학적 데이터를 분석하는 방법을 제공한다. 상기 방법은 곡선의 한 세그먼트와 방향에 의해 정의되는 한 축 사이의 거리를 계산하는 단계를 포함하며, 상기 거리는 상기 방향에 따른 좌표 δ1에 의해 정의되는 곡선 위의 한 점에서 계산된다. 상기 방법은 상기 거리를 대상체의 박테리아에 감염 여부, 또는 박테리아 감염 가능성과 상관시키는 단계를 더 포함한다. 좌표 δ1은 발현값의 조합에 의해 정의되며, 세그먼트의 적어도 90 %는 하한선 f(δ1)-ε0와 상한선 f(δ1)+ε1 사이에 있고, f(δ1)은 1/(1+exp(δ1))과 동일하며, ε0 및 ε1 각각은 0.5 미만이다.
본 발명의 일부 실시예에 따르면, 상기 방법은 상기 거리에 기초하여 가능성를 얻는 단계, 상기 가능성을 미리 결정된 임계값과 비교하는 단계, 및 상기 가능성이 상기 미리 결정된 임계값을 초과하는 경우 상기 박테리아성 감염 대상체를 치료하는 단계를 포함한다.
본 발명의 일부 실시예에 따르면, 대상체의 혈액 중의 복수의 폴리펩타이드의 발현값을 포함하는 생물학적 데이터를 분석하는 방법을 제공한다. 상기 방법은 곡선의 한 세그먼트와 방향에 의해 정의된 한 축 사이의 거리를 계산하는 단계를 포함하며, 상기 거리는 상기 방향을 따른 좌표 δ0에 의해 정의되는 곡선 위의 한 점에서 계산된다. 이 방법은 상기 거리를 대상체의 바이러스성 감염 여부 또는 바이러스성 감염 가능성과 상관시키는 단계를 더 포함한다. 좌표 δ0은 발현값의 조합에 의해 정의되며, 세그먼트의 적어도 90 %는 하한선 g(δ0)-ε0와 상한선 g(δ0)+ε1 사이에 있고, g(δ0)은 1/(1+exp(δ0))과 동일하며, ε0 및 ε1 각각은 0.5 미만이다.
본 발명의 일부 실시예에 따르면, 상기 방법은 상기 거리에 기초하여 가능성을 얻는 단계, 상기 가능성을 미리 결정된 임계값과 비교하는 단계, 및 상기 가능성이 상기 미리 결정된 임계값을 초과하는 경우 상기 바이러스성 감염 환자를 치료하는 단계를 포함한다.
본 발명의 일부 실시예에 따르면, 발현값의 조합은 발현값의 선형 결합(a linear combination)을 포함한다.
본 발명의 일부 실시예에 따르면, 발현값의 조합은 발현값 중 적어도 하나에 대응하는 적어도 하나의 비선형항(nonlinear term)을 포함한다.
본 발명의 일부 실시예에 따르면, 대상체의 혈액 중의 복수의 폴리펩타이드의 발현값을 포함하는 생물학적 데이터를 분석하는 방법을 제공한다. 상기 방법은 곡면의 한 세그먼트와 제 1 방향 및 제 2 방향에 의해 정의되는 평면 사이의 제 1 거리를 계산하는 단계를 포함한다. 제 1 거리는 제 1 방향을 따른 제 1 좌표 δ0 및 제 2 방향을 따른 제 2 좌표 δ1에 의해 정의된 평면상의 점에서 계산된다. 이 방법은 제 1 거리를 대상체의 박테리아성 감염 여부 또는 박테리아성 감염 가능성과 상관시키는 단계를 더 포함한다. 각각의 좌표는 발현값의 다른 조합에 의해 정의되며, 세그먼트의 적어도 90 %는 하한면 f(δ0,δ1)-ε0와 상한면 f(δ0,δ1)+ε1 사이에 있고, f(δ0,δ1)은 exp(δ1)/(1+exp(δ0)+exp(δ1))과 동일하며, ε0 및 ε1 각각은 0.5 미만이다.
좌표 중 적어도 하나에 대한 본 발명의 일부 실시예에 따르면, 발현 값의 조합은 발현값의 선형 결합을 포함한다.
좌표 중 적어도 하나에 대한 본 발명의 일부 실시예에 따르면, 발현값의 조합은 발현값 중 적어도 하나에 대응하는 적어도 하나의 비선형항(nonlinear term)을 포함한다.
본 발명의 일부 실시예에 따르면, 상기 방법은 상기 거리에 기초하여 가능성을 얻는 단계, 상기 가능성을 미리 결정된 임계값과 비교하는 단계, 및 상기 가능성이 상기 미리 결정된 임계값을 초과하는 경우 상기 박테리아성 감염 환자를 치료하는 단계를 포함한다.
본 발명의 일부 실시예에 따르면, 상기 방법은 제 2 곡면의 세그먼트와 상기 평면 사이의 제 2 거리를 계산하는 단계; 및 제 2 거리를 대상체의 바이러스성 감염 여부 또는 바이러스성 감염 가능성과 상관시키는 단계를 포함한다. 본 발명의 일부 실시예에 따르면, 제 2 곡면의 세그먼트의 적어도 90 %는 하한면 g(δ0,δ1)-ε2와 상한면 g(δ0,δ1)+ε3 사이에 있고, g(δ0,δ1)은 exp(δ0)/(1+exp(δ0)+exp(δ1))과 동일하며, ε2 및 ε3 각각은 0.5 미만이다.
본 발명의 일부 실시예에 따르면, 상기 방법은 상기 거리에 기초하여 가능성을 얻는 단계, 상기 가능성을 미리 결정된 임계값과 비교하는 단계, 및 상기 가능성이 상기 미리 결정된 임계값을 초과하는 경우 상기 바이러스성 감염 환자를 치료하는 단계를 포함한다.
본 발명의 일부 실시예에 따르면, 상기 방법은 상기 거리에 기초하여 상기 다상체의 박테리아성 감염 가능성을 얻는 단계, 상기 제 2 거리에 기초하여 상기 대상체의 바이러스성 감염 가능성을 얻는 단계, 상기 가능성의 각각을 미리 결정된 임계값과 비교하는 단계 가능성의 각각이 각각의 미리 결정된 임계값 이하일 때, 대상체가 비-감염성 질병을 가질 가능성이 있다고 결정하는 단계를 포함한다.
본 발명의 일부 실시예에 따르면, 대상체의 혈액 중의 복수의 폴리펩타이드의 발현값을 포함하는 생물학적 데이터를 분석하는 방법을 제공한다. 상기 방법은 곡면의 한 세그먼트와 제 1 방향 및 제 2 방향으로 정의된 평면 사이의 거리를 계산하는 단계를 포함한다. 상기 거리는 제 1 방향을 따른 제 1 좌표 δ0 및 제 2 방향을 따른 제 2 좌표 δ1에 의해 정의된 평면 위의 한 점에서 계산된다. 상기 방법은 상기 거리를 대상체의 바이러스성 감염 여부 또는 감염 가능성과 상관시키는 단계를 포함한다. 상기 각가의 좌표는 발현값의 상이한 조합에 의해 정의되고, 상기 세그먼트의 적어도 90 %는 하한면 g(δ0,δ1)-ε0와 상한면 g(δ0,δ1)+ε1사이에 존재하고, 상기 g(δ0,δ1)은 exp(δ0)/(1+exp(δ0)+exp(δ1))와 동일하며, ε0 및 ε1 각각은 0.5 미만이다.
본 발명의 일부 실시예에 따르면, 복수의 폴리펩타이드 각각은 CRP, IP-10, TRAIL, IL1ra, PCT 및 SAA로 이루어진 군으로부터 선택된다.
본 발명의 일부 실시예에 따르면, 복수의 폴리펩타이드는 적어도 3 개의 폴리펩타이드를 포함한다.
본 발명의 일부 실시예에 따르면, 복수의 폴리펩타이드는 CRP, IP-10, TRAIL, IL1ra, PCT 및 SAA로 이루어진 군으로부터 선택된 3종 이상의 폴리펩타이드를 포함한다.
본 발명의 일부 실시예에 따르면, 복수의 폴리펩타이드는 적어도 CRP 및 TRAIL을 포함한다.
본 발명의 일부 실시예에 따르면, 복수의 폴리펩타이드는 적어도 CRP, TRAIL 및 IP-10을 포함한다.
본 발명의 일부 실시예에 따르면, 상기 방법은 가능성의 결과를 생성하는 단계를 포함하고, 상기 결과는 텍스트로 나타낸다.
본 발명의 일부 실시예에 따르면, 상기 방법은 가능성의 결과를 생성하는 단계를 포함하고, 상기 결과는 그래프로 나타낸다.
본 발명의 일부 실시예에 따르면, 상기 방법은 가능성의 결과를 생성하는 단계를 포함하고, 상기 결과는 컬러 인덱스를 사용하여 나타낸다.
본 발명의 일부 실시예에 따르면, 상기 혈액 샘플은 전혈이다.
본 발명의 일부 실시예에 따르면,, 상기 혈액 샘플은 전혈의 분획이다.
본 발명의 일부 실시예에 따르면, 상기 혈액의 분획은 혈청 또는 혈장이다.
본 발명의 일부 실시예에 따르면, 상기 방법은 발현값을 결정하는 단계를 포함하고, 상기 발현값 중 하나 이상은 전기영동적으로 또는 면역화학적으로 결정된다.
본 발명의 일부 실시예에 따르면, 상기 면역화학적 결정은 유세포 분석법(flow cytometry), 방사 면역 분석법(radioimmunoassay), 면역 형광법(immunofluorescence) 또는 효소 결합 면역 흡착 분석법(enzyme-linked immunosorbent assay)에 의해 수행된다.
본 발명의 일부 실시예에 따르면, 계산하고 상관시키는 단계는 대상체로부터 떨어진 컴퓨터에 의해 실행된다.
본 발명의 일부 실시예에 따르면, 상기 상기 계산하는 단계 및 연관시키는 단계는 대상체에 근접한 컴퓨터에 의해 실행된다.
본 발명의 일부 실시예에 따르면, 상기 계산하는 단계 및 연관시키는 단계는 클라우드 컴퓨터 설비의 클라우드 컴퓨터 리소스에 의해 실행된다.
본 발명의 일부 실시예에 따르면, 발현값은 자동화된 ELISA, 자동화된 면역 분석 및 자동화된 기능 분석으로 이루어진 군으로부터 선택된 하나 이상의 자동화된 분석법을 수행하는 측정 시스템에 의해 측정되며, 상기 방법은 상기 측정 시스템으로부터 생물학적 데이터를 얻는 단계를 포함한다.
본 발명의 일부 실시예에 따르면, 상기 생물학적 데이터를 얻는 단계는 네트워크 인터페이스에 의한 인터넷 네트워크를 통해 이루어진다.
본 발명의 일부 실시예의 일 측면에 따르면, 생물학적 데이터를 분석하기 위한 컴퓨터 구현 방법을 제공한다. 상기 방법은, 계산 활성화 제어를 갖는 그래픽 사용자 인터페이스 (GUI)를 디스플레이 장치 상에 디스플레이하는 단계; 대상체의 혈액 중 폴리펩타이드의 발현값을 얻는 단계; 사용자에 의한 활성화 제어에 응답하여 상기 발현값에 기초하여 점수를 자동으로 계산하는 단계; 대상체의 바이러스성 감염에 상응하는 것으로 식별된 제 1 단부 및 대상체의 박테리아성 감염에 상응하는 것으로 식별된 제 2 단부를 갖는 그래픽 스케일을 GUI 상에 생성하는 단계; 및 상기 점수에 상응하는 위치에서 상기 스케일 상에 마크를 생성하는 단계를 포함한다.
본 발명의 일부 실시예에 따르면, 발현값은 발현값을 측정하는 외부 기계와 통신함으로써 얻는다. 본 발명의 일부 실시예에 따르면, GUI는 통신 제어를 포함하며, 여기서 외부 기계와의 통신은 사용자에 의한 통신 제어의 활성화에 반응하는 것이다.
본 발명의 일부 실시예에 따르면, GUI는 복수의 발현값 입력 필드를 포함하며, 발현값은 입력 필드를 통해 얻는다.
본 발명의 일부 실시예에 따르면, 점수는 대상체의 박테리아성 감염 가능성을 나타낸다. 본 발명의 일부 실시예에 따르면, 점수는 대상체의 바이러스성 감염 가능성을 나타낸다.
본 발명의 일부 실시예에 따르면, 프로그램 명령어가 저장되는 컴퓨터 판독 가능 매체를 포함하고, 상기 명령어는 하드웨어 프로세서에 의해 판독될 때 상기 하드웨어 프로세스로 하여금 미지의 질병을 가진 대상체의 혈액 중의 다수의 폴리펩타이드의 발현값을 얻도록 하고, 그리고 상기 기술되고 선택적으로 하기에 더 상세히 설명되는 바와 같은 방법 실행되는 컴퓨터 소프트웨어 제품을 제공한다.
본 발명의 일부 실시예에 따르면, 생물학적 데이터를 분석하는 시스템이 제공된다. 이 시스템은 미지의 질병을 가진 대상체의 혈액에서 복수의 폴리펩타이드의 발현값을 얻도록 구성된 사용자 인터페이스; 및 상기 컴퓨터 소프트웨어 제품을 저장하는 컴퓨터 판독 가능 매체를 갖는 하드웨어 프로세서를 포함하는 컴퓨터 판독 가능 저장 매체를 포함한다.
본 발명의 일부 실시예에 따르면, 본 발명은 생물학적 데이터를 분석하는 시스템을 제공한다. 상기 시스템은 미지의 질병을 가진 대상체의 혈액에서 복수의 폴리펩타이드의 발현값을 측정하도록 구성된 제 1 구획; 컴퓨터 소프트웨어 제품을 저장하는 컴퓨터 판독 기능을 갖는 하드웨어 프로세서를 포함하는 제 2 구획을 포함한다.
따르면, 제 1 구획, 제 2 구획 및 디스플레이는 휴대용 장치(hand-held device)의 본체 상에 장착되거나 또는 일체화된다.
본 발명의 일부 실시예의 한 측면에 따르면, 본 발명은 데이터 세트를 분석하는 방법을 제공한다. 이 방법은 (a) 다수의 대상체의 혈액 샘플에서 미지의 질병을 갖는 대상체의 혈액에서 복수의 폴리펩타이드의 발현값에 기초한 분류 그룹을 포함하는 데이터 세트에 엑세스하는 단계 - 상기 분류 그룹은 박테리아성 감염, 바이러스성 감염 및 비-바이러스성, 비-박테리아성 질병; 및 (b) 특정 대상체의 박테리아성 감염 가능성을 나타내는 적어도 제 1 확률 분류 함수 f(δ0,δ1)를 제공하도록 분류 그룹을 분석하는 단계로서, 상기 제 1 분류 함수는 제 1 좌표 δ0 및 제 2 좌표 δ1의 함수이고, 상기 좌표들 각각은 상기 발현값들의 상이한 조합에 의해 정의되는 것을 특징으로 하는 단계를 포함한다.
본 발명의 일부 실시예에 따르면, 상기 방법은 특정 대상체의 바이러스성 감염 가능성을 나타내는 제 2 분류 함수 g(δ0,δ1)를 계산하는 단계를 더 포함하며, 상기 제 2 분류 함수는 또한 상기 제 1 및 제 2 좌표의 함수이다.
본 발명의 일부 실시예에 따르면, 상기 방법은 특정 대상체의 비 바이러스성, 비-박테리아성 질병을 가질 가능성을 나타내는 제 3 분류 함수 h(δ0,δ1)를 계산하는 단계를 포함하며, 상기 제 3 분류 함수는 또한 상기 제 1 및 제 2 좌표의 함수이다.
본 발명의 일부 실시예에 따르면, 상기 좌표들 중 적어도 하나에 대해, 발현값의 조합은 발현값의 선형 결합을 포함한다.
좌표 중 적어도 하나에 대한 본 발명의 일부 실시예에 따르면, 발현값의 조합은 발현값 중 적어도 하나에 대응하는 적어도 하나의 비선형항을 포함한다.
본 발명의 일부 실시예에 따르면, 상기 방법은 상기 분석의 결과를 생성하는 단계를 포함한다.
본 발명의 일부 실시예에 따르면, 데이터 세트는 하나 이상의 다차원 항목을 포함한다.
본 발명의 일부 실시예에 따르면, 데이터 세트의 각 항목은 각각의 대상체의 적어도 하나의 임상 파라미터를 포함한다.
본 발명의 일부 실시예에 따르면, 임상 파라미터는 성별, 연령, 온도, 발병기간 및 체중으로 이루어진 군으로부터 선택된다.
본 발명의 일부 실시예에 따르면, 분석은 머신 러닝을 포함한다.
본 발명의 일부 실시예에 따르면, 머신 러닝은 지도받는 머신 러닝을 포함한다.
본 발명의 일부 실시예에 따르면, 상기 머신 러닝은 클러스터링(clustering), 서포트 벡터 머신(support vector machine), 선형 모델링(linear modeling), k-니어리스트 네이버 분석(k-nearest neighbors analysis), 결정 트리 러닝(decision tree learning), 앙상블 러닝 절차(ensemble learning procedure), 신경 네트워크(neural networks), 확률 모델(probabilistic model), 그래픽 모델(graphical model), 베이지안 네트워크(Bayesian network) 로지스틱 회귀(logistic regression) 및 협회 규칙 러닝(association rule learning)으로 이루어진 군으로부터 선택되는 하나 이상의 절차를 포함한다.
본 발명의 일부 실시예에 따르면, 상기 머신 러닝은 서포트 벡터 머신, 신경 네트워크 및 로지스틱 회귀로 이루어진 군으로부터 선택된다.
본 발명의 일부 실시예에 따르면, 상기 혈액 샘플은 전혈이다.
본 발명의 일부 실시예에 따르면, 상기 혈액 샘플은 전혈의 분획이다.
본 발명의 일부 실시예에 따르면, 상기 혈액의 분획은 혈청 또는 혈장을 포함한다.
본 발명의 일부 실시예에 따르면, 상기 발현값은 전기영동적으로 또는 면역화학적으로 결정된다.
본 발명의 일부 실시예에 따르면, 면역화학적 결정은 유세포 분석법, 방사 면역 분석법, 면역 형광법 또는 효소 결합 면역 흡착 분석법에 의해 수행된다.
본 발명의 일부 실시예에 따르면, 질병의 예후를 예측하는 방법을 제공한다. 상기 방법은 상기 질병을 갖는 대상체에서 TRAIL 단백질 혈청 레벨을 측정하는 단계를 포함하고, 상기 TRAIL 레벨이 미리 결정된 레벨보다 이하인 경우, 상기 예후는 상기 미리 결정된 레벨을 초과하는 TRAIL 단백질 혈청 레벨을 갖는 질병을 갖는 대상체보다 악화된 것이다.
본 발명의 일부 실시예에 따르면, 상기 질병은 감염성 질병이다.
본 발명의 일부 실시예에 따르면, 상기 질병은 감염성 질병이 아니다.
본 발명의 일부 실시예에 따르면, 대상체 내 질병에 대한 치료 과정을 결정하는 방법을 제공한다. 상기 방법은 대상체 내 TRAIL 단백질 혈청 레벨을 측정하는 단계를 포함하고, 상기 TRAIL 레벨이 미리 결정된 레벨 이하인 경우, 상기 대상체는 최후의 수단으로 치료받게 된다.
본 발명의 일부 실시예에 따르면, 상기 미리 결정된 레벨은 20 pg/ml 이하이다.
본 발명의 일부 실시예의 한 측면에 따르면, 본 발명은 임신 가능한 여성 대상체 내 감염 유형을 결정하는 방법을 제공한다.
상기 방법은 대상체의 TRAIL 단백질 혈청 레벨을 임신 가능한 건강한 여성 또는 임신 가능한 건강한 여성 대상체 그룹의 TRAIL 단백질 혈청 레벨에 상응하는 미리 결정된 임계값과 비교하는 단계를 포함하고, 상기 TRAIL 단백질 혈청 레벨과 상기 미리 결정된 임계값 간의 차이가 감염 유형을 나타내다.
본 발명의 일부 실시예의 한 측면에 따르면, 본 발명은 임신 가능한 남성 대상체 내 감염 유형을 결정하는 방법을 제공한다.
상기 방법은 대상체의 TRAIL 단백질 혈청 레벨을 임신 가능한 건강한 남성 또는 임신 가능한 건강한 남성 대상체 그룹의 TRAIL 단백질 혈청 레벨에 상응하는 미리 결정된 임계값과 비교하는 단계를 포함하고, 상기 TRAIL 단백질 혈청 레벨과 상기 미리 결정된 임계값 간의 차이가 감염 유형을 나타내다.
본 발명의 일부 실시예에 따르면, 상기 TRAIL 단백질 혈청 레벨이 미리 결정된 레벨 이상인 경우, 감염 유형은 바이러스성이다.
본 발명의 일부 실시예에 따르면, 상기 TRAIL 단백질 혈청 레벨이 미리 결정된 레벨 이상인 경우, 감염 유형은 박테리아성이다.
본 발명의 일부 실시예에 따르면, 상기 TRAIL 단백질 혈청 레벨이 미리 결정된 레벨 이하인 경우, 감염 유형은 박테리아성이다.
본 발명의 일부 실시예에 따르면, 상기 TRAIL 단백질 혈청 레벨이 미리 결정된 레벨 이하인 경우, 감염 유형은 바이러스성이 아니다.
달리 정의되지 않는 한, 본 명세서에서 사용된 모든 기술 및/또는 과학 용어는 본 발명이 속하는 기술 분야의 통상의 기술자에 의해 일반적으로 이해되는 것과 동일한 의미를 갖는다. 본 명세서에 기술된 것들과 유사하거나 동등한 방법 및 재료가 본 발명의 실시예를 실행 또는 테스트하는데 사용될 수 있지만, 예시적인 방법 및/또는 재료는 이하 기술된다. 모호한 경우, 정의를 포함한 특허 명세서가 우선한다. 또한, 재료, 방법 및 실시예는 단지 예시적인 것이며 반드시 제한하려는 것은 아니다.
본 발명의 일부 실시예는 첨부 도면을 참조하여 단지 예시적으로 설명한다. 이제 도면을 상세하게 구체적으로 참조하면, 도시된 세부 사항은 예시로서 그리고 본 발명의 실시예에 대한 설명을 목적으로 한 것임을 강조한다. 이와 관련하여, 도면과 함께 기술된 설명은 본 발명의 실시예가 실시될 수 있는 방법을 통상의 기술자에게 명백하게 한다.
도 1a-1b. 연구 워크플로우. (1a) 연구 워크플로우의 개요. n박테리아성, n바이러스성 및 n대조군 은 박테리아성(혼합된 박테리아성 및 바이러스성 공동 감염 포함), 바이러스성 및 대조군(감염성 질병 없음)을 나타낸다. (1b) 단백질 발견 및 검증 과정.
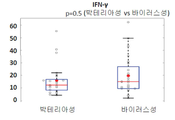
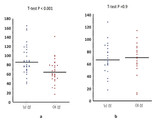
도 2a-2c. 단백질 TRAIL, IP-10 및 CRP는 박테리아성, 바이러스성 및 비-감염성 환자들에서 다르게 발현된다. Majority cohort (n=765)에 대해 측정된 TRAIL (2a), IP-10 (2b) 및 CRP (2c)에 대한 박스 플랏(Box plots)을 나타내었다. 빨간선 및 원은 각각 그룹 평균 및 평균에 해당하고; 박테리아성 및 바이러스성 그룹 사이와 감염성(박테리아성 및 바이러스성) 및 비-감염성(건강한 대상체 포함) 사이의 t-test p-values를 나타내었다.
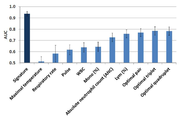
도 3a-3b. 박테리아성 대 바이러스성 환자를 진단하기 위한 시그니쳐와 실험 파라미터 및 단백질 바이오마커와의 비교. (3a) 실험 파라미터 뿐만 아니라 가장 우수한 성능을 보이는 파라미터들의 2중 조합 (ANC 및 Lym %), 3중 조합(ANC, Lym % 및 Pulse) 및 4중 조합 (ANC, Lym %, Pulse 및 Mono %)의 임상적 성능으로, 상기 값들은 로지스틱 회귀(logistic regression)를 사용하여 결합되었다. 맥박 (박테리아성 환자 292명, 바이러스성 환자 326명) 및 호흡률 (박테리아성 환자 292명, 바이러스성 환자 326명)과 별개로 대다수 집단 (박테리아성 및 바이러스성 환자, n = 653)에 대한 비교가 이루어졌다. 상기 시그니쳐는 최적의 4중 조합보다 상당히 우수하였다 (P<10-15). (3b) 상기 시그니쳐는 감염에 대한 숙주 반응에서 잘 확립된 역할을 하는 바이오마커들 보다 상당히 우수하였다 (P<10-8). 각각의 선택 바이오마커에 대해, 분석은 대다수 집단(각 분석에 대해 43 ≤ n ≤154, 편의 샘플, n은 신호의 세기에 의존)의 하위 그룹에서 수행되었다. 오차 바(Error bars)는 95% CI를 나타낸다.
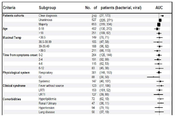
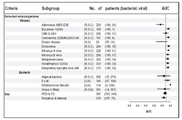
도 4. 시그니쳐 성능은 서로 다른 환자 하위그룹에서 강하다. 대다수 집단(박테리아성 및 바이러스성)의 하위그룹에서 시그니쳐 AUC를 도시하였다. 사각형 크기는 환자 수에 비례하며 오차 바는 95% CI를 나타낸다. 병원균 분석에서, 각 바이러스성를 괄호 안에 표시된 동일한 생리학적 시스템에 영향을 미치는 박테리아성와 비교하였다. R- 호흡기, S- 전신, C- 중추 신경계, G- 위장관, U- 비뇨기, K- 피부. 5명 이상의 환자에서 검출된 병원균만을 나타낸다. 하위그룹 정의에 대해서는 실시예1의 표 1을 참고한다.
도 5. MLR 모델의 눈금 플랏(Calibration plot). 상부 패널에서 환자들을 예측된 박테이라성 감염(x축) 확률을 기초로 10개의 빈(bin)으로 그룹화하고, 각 빈 내의 관찰된 박테리아성 감염률(y축)을 비교하였다. 점선은 이동 평균(5개 빈 크기)이다. 하부 패널은 박테리아성(빨간 막대) 및 바이러스성 (파란 막대)에 대한 예상 확률 분포를 나타낸다.
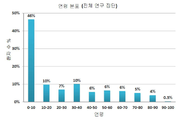
도 6a-6b. 진단 환자들의 연령 분포. 6a. 전체 연구 인원 (n=794); 6b. 소아 환자만 (n=445).
도 7a-7b. 진단 환자(n=794)에서 관찰된 병원균의 분포. 7a. 병원성 하위그룹에 의해 검출된 병원균의 분포; 7b. 균주(환자의 >1% 으로부터 검출된 균주들)에 의해 검출된 병원균의 분포. 분포는 진단된 감염성 질병을 가진 환자들에서 양성 검출의 백분율을 나타낸다.
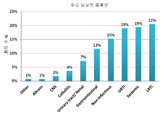
도 8. 감염성 질병을 가진 것으로 진단된 환자(n=673)에서 관련된 생리학적 시스템의 분포.
도 9a-9b. 임상적 증후군(모든 진단 환자, n=794)의 분포. 9a. 주요 임상적 증후군; 9b. 특이적 임상적 증후군.
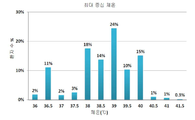
도 10. 최대 체온 분포(n=794).
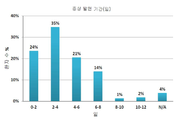
도 11. 증상이 발생한 기간의 분포(n=794). N/A- 데이터를 얻지 못한 건강한 대조군 또는 환자.
도 12a-12b. 환자 집단의 합병증과 관련된 특성. 12a. 합병증의 분포(모든 만성 질환 환자. n=305); 12b. 만성 약물의 분포(모든 만성 질환 환자. n=305). 일부 환자들은 여러 만성 질병을 가지고, 여러 만성 약물로 치료받은 것으로 나타났다.
도 13. 모집군 분포 (진단 환자, n=794)
도 14a-14b. 시그니쳐에 대한 추정 PPV 및 NPV 값 박테리아성 감염 유병률의 함수, 14a. 만장일치 (박테리아성, 바이러스성) 집단 (n=527), 14b. 대다수 (박테리아성, 바이러스성) 집단 (n=653).
도 15a-15b. 대다수 (박테리아성, 바이러스성, 비-감염성) 집단 (n=765)에서 박테리아성, 바이러스성 및 비-감염성 환자들에서의 임상 파라미터 및 실험실 측정의 산탄도. 빨간선 및 원은 각각 그룹 중앙값과 평균에 해당한다. 박테리아성 및 바이러스성 그룹간과 감염성(박테리아성 및 바이러스성) 및 비-감염성(건강한 대상체 포함)간의 T-test p-values를 도시했다.
도 16a-16b. 서로 다른 컷오프를 사용하여 시그니쳐 및 PCT의 성능을 비교. 16a. 만장일치 (박테리아성, 바이러스성) 집단으로부터 76 환자에서 측정된 성능; 16b 대다수 (박테리아성, 바이러스성) 집단으로부터 101 환자에서 측정된 성능. 오류 바는 95% CI를 나타낸다. 시그니쳐 민감도 및 특이성을 한계 면역 반응을 하는 환자의 14%를 필터링한 후 계산하였다.
도 17a-17b. 서로 다른 컷오프를 사용하여 시그니쳐 및 CRP의 성능을 비교. 17a. 만장일치 (박테리아성, 바이러스성) 집단(n=527)으로부터 76 환자에서 측정된 성능; 17b. 대다수 (박테리아성, 바이러스성) 집단 (n=653)로부터 101 환자에서 측정된 성능. 오류 바는 95% CI를 나타낸다. 시그니쳐 민감도 및 특이성을 한계 면역 반응을 하는 환자의 14%를 필터링한 후 계산하였다.
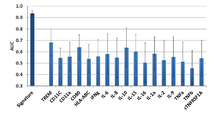
도 18a-18h. 박테리아성 및 바이러스성 환자들에서 선택된 단백질 바이오마커(임의 단위)의 레벨의 산탄도. 빨간선 및 원은 각각 중앙값과 평균에 해당한다. 박테리아성 및 바이러스성 그룹간의 T-test p-values을 도시했다.
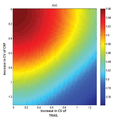
도 19a-19b. 시그니쳐의 임상적 정확성은 단백질 측정의 기술적 정확성에서의 감소에 강하다. (19a) 바이러스성 감염으로부터 박테리아성을 구별하는 시그니쳐의 AUC를 TRAIL (y축) 및 CRP (x축) 측정의 CV(std/mean) 함수로써 컬러맵을 사용하여 측정하였다. (19b)도 19A의 대각선 상의 AUC 값은 TRAIL 및 CRP의 CV가 동일하게 표시 되었다.
도 20은 박테리아성 (‘+’), 바이러스성 (‘o’) 및 비-감염성 (‘^’) 환자들의 3차원 시각화이다. 다른 환자들 유형이 CRP (μg/ml), TRAIL 및 IP-10 (pg/ml) 농도 맵에서 구별되는 영역으로 맵핑된다.
도 21a-21c. 0-100의 IP-10 범위에 대한 본 발명의 일부 실시예에 따라 얻어진 것과 같이, TRAIL (y축), CRP (x축) 및 IP-10 농도의 함수로서 바이러스성(21a), 박테리아성 또는 혼합된(21b) 및 비-감염성 또는 건강한(21c)의 확률.
도 22a-22c. 100-200의 IP-10 범위에 대한 본 발명의 일부 실시예에 따라 얻어진 것과 같이, TRAIL (y축), CRP (x축) 및 IP-10 농도의 함수로서 바이러스성(22a), 박테리아성 또는 혼합된(22b) 및 비-감염성 또는 건강한(22c)의 확률.
도 23a-23c. 200-300의 IP-10 범위에 대한 본 발명의 일부 실시예에 따라 얻어진 것과 같이, TRAIL (y축), CRP (x축) 및 IP-10 농도의 함수로서 바이러스성(23a), 박테리아성 또는 혼합된(23b) 및 비-감염성 또는 건강한(23c)의 확률.
도 24a-24c. 300-400의 IP-10 범위에 대한 본 발명의 일부 실시예에 따라 얻어진 것과 같이, TRAIL (y축), CRP (x축) 및 IP-10 농도의 함수로서 바이러스성(24a), 박테리아성 또는 혼합된(24b) 및 비-감염성 또는 건강한(24c)의 확률.
도 25a-25c. 400-500의 IP-10 범위에 대한 본 발명의 일부 실시예에 따라 얻어진 것과 같이, TRAIL (y축), CRP (x축) 및 IP-10 농도의 함수로서 바이러스성(25a), 박테리아성 또는 혼합된(25b) 및 비-감염성 또는 건강한(25c)의 확률.
도 26a-26c. 500-600의 IP-10 범위에 대한 본 발명의 일부 실시예에 따라 얻어진 것과 같이, TRAIL (y축), CRP (x축) 및 IP-10 농도의 함수로서 바이러스성(26a), 박테리아성 또는 혼합된(26b) 및 비-감염성 또는 건강한(26c)의 확률.
도 27a-27c 1000-2000의 IP-10 범위에 대한 본 발명의 일부 실시예에 따라 얻어진 것과 같이, TRAIL (y축), CRP (x축) 및 IP-10 농도의 함수로서 바이러스성(27a), 박테리아성 또는 혼합된(27b) 및 비-감염성 또는 건강한(27c)의 확률.
도 28a-28c. 2000 이상의 IP-10 범위에 대한 본 발명의 일부 실시예에 따라 얻어진 것과 같이, TRAIL (y축), CRP (x축) 및 IP-10 농도의 함수로서 바이러스성(28a), 박테리아성 또는 혼합된(28b) 및 비-감염성 또는 건강한(28c)의 확률.
도 29a-29f. 본 발명의 일 실시예에 따른 박테리아성 및 비-박테리아성 감염을 구별하는 방법의 예시적인 결과.
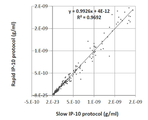
도 30a-30b. TRAIL (도 30a) 및 IP-10 (도 30b)의 측정을 위한 빠르고 느린 프로토콜 간의 상관 관계를 나타내는 그래프.
도 31. 본 발명의 다양한 예시적 실시예에 따라 대상체로부터 얻은 생물학적 데이터를 분석하는 적합한 방법의 흐름도.
도 32a-32b. 본 발명의 일부 실시예에 따른 한 평면으로부터 한 표면의 거리를 계산하는 절차를 설명하는 개략도.
도 33a-33d. 본 발명의 일부 실시예에 따른 표면의 한 부드러운 세그먼트를 얻기 위한 절차를 설명하는 개략도.
도 34. 본 발명의 일부 실시예에 따라 생물학적 데이터를 분석하는 시스템의 블록 개략도.
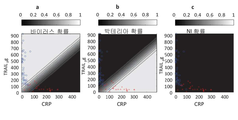
도 35a-35d. 박테리아성 (도 35a), 바이러스성 (도 35b), 비-박테리아성 (도 35c) 및 비-감염성 (도 35d) 병인의 확률을 좌표 δ0 및 δ1의 함수로서 설명하는 등고선 플롯. 상기 확률값 범위는 0% (검은색) to 100% (흰색) 사이이다.
도 36a-36b. 낮은 TRAIL 레벨은 환자의 예후와 결과가 좋지 않고 질병의 심각도가 높다는 표지이다. (36a) 다른 모든 환자(감염성 또는 비-감염성 병인이 있는)와 비교한 ICU에 입원한 환자의 혈청 내 TRAIL 농도. (36b) 감염성 또는 비-감염성 병인이 있는 다른 모든 환자와 비교하여 ICU에 입원했거나 사망한 소아 환자의 혈청 내 TRAIL 농도.
도 37a-37b. 임신 가능한 연령의 남성 및 여성 내 TRAIL 농도 차이를 나타낸 그래프.
도 38a-38e. 본 발명의 일부 실시예에 따라 생물학적 데이터를 분석하기 위한 컴퓨터 구현 방법에서 사용자 입력을 수신하는데 적합한 그래픽 사용자 인터페이스 (GUI)의 스크린 샷.
도 39a-39b. 본 발명의 실시예에서, 네트워크 인터페이스 (도 39a) 및 사용자 인터페이스 (도 39b)를 포함하는 시스템에서 생물학적 데이터를 분석하기 위한 시스템의 블록 개략도.
도 1a-1b. 연구 워크플로우. (1a) 연구 워크플로우의 개요. n박테리아성, n바이러스성 및 n대조군 은 박테리아성(혼합된 박테리아성 및 바이러스성 공동 감염 포함), 바이러스성 및 대조군(감염성 질병 없음)을 나타낸다. (1b) 단백질 발견 및 검증 과정.
도 2a-2c. 단백질 TRAIL, IP-10 및 CRP는 박테리아성, 바이러스성 및 비-감염성 환자들에서 다르게 발현된다. Majority cohort (n=765)에 대해 측정된 TRAIL (2a), IP-10 (2b) 및 CRP (2c)에 대한 박스 플랏(Box plots)을 나타내었다. 빨간선 및 원은 각각 그룹 평균 및 평균에 해당하고; 박테리아성 및 바이러스성 그룹 사이와 감염성(박테리아성 및 바이러스성) 및 비-감염성(건강한 대상체 포함) 사이의 t-test p-values를 나타내었다.
도 3a-3b. 박테리아성 대 바이러스성 환자를 진단하기 위한 시그니쳐와 실험 파라미터 및 단백질 바이오마커와의 비교. (3a) 실험 파라미터 뿐만 아니라 가장 우수한 성능을 보이는 파라미터들의 2중 조합 (ANC 및 Lym %), 3중 조합(ANC, Lym % 및 Pulse) 및 4중 조합 (ANC, Lym %, Pulse 및 Mono %)의 임상적 성능으로, 상기 값들은 로지스틱 회귀(logistic regression)를 사용하여 결합되었다. 맥박 (박테리아성 환자 292명, 바이러스성 환자 326명) 및 호흡률 (박테리아성 환자 292명, 바이러스성 환자 326명)과 별개로 대다수 집단 (박테리아성 및 바이러스성 환자, n = 653)에 대한 비교가 이루어졌다. 상기 시그니쳐는 최적의 4중 조합보다 상당히 우수하였다 (P<10-15). (3b) 상기 시그니쳐는 감염에 대한 숙주 반응에서 잘 확립된 역할을 하는 바이오마커들 보다 상당히 우수하였다 (P<10-8). 각각의 선택 바이오마커에 대해, 분석은 대다수 집단(각 분석에 대해 43 ≤ n ≤154, 편의 샘플, n은 신호의 세기에 의존)의 하위 그룹에서 수행되었다. 오차 바(Error bars)는 95% CI를 나타낸다.
도 4. 시그니쳐 성능은 서로 다른 환자 하위그룹에서 강하다. 대다수 집단(박테리아성 및 바이러스성)의 하위그룹에서 시그니쳐 AUC를 도시하였다. 사각형 크기는 환자 수에 비례하며 오차 바는 95% CI를 나타낸다. 병원균 분석에서, 각 바이러스성를 괄호 안에 표시된 동일한 생리학적 시스템에 영향을 미치는 박테리아성와 비교하였다. R- 호흡기, S- 전신, C- 중추 신경계, G- 위장관, U- 비뇨기, K- 피부. 5명 이상의 환자에서 검출된 병원균만을 나타낸다. 하위그룹 정의에 대해서는 실시예1의 표 1을 참고한다.
도 5. MLR 모델의 눈금 플랏(Calibration plot). 상부 패널에서 환자들을 예측된 박테이라성 감염(x축) 확률을 기초로 10개의 빈(bin)으로 그룹화하고, 각 빈 내의 관찰된 박테리아성 감염률(y축)을 비교하였다. 점선은 이동 평균(5개 빈 크기)이다. 하부 패널은 박테리아성(빨간 막대) 및 바이러스성 (파란 막대)에 대한 예상 확률 분포를 나타낸다.
도 6a-6b. 진단 환자들의 연령 분포. 6a. 전체 연구 인원 (n=794); 6b. 소아 환자만 (n=445).
도 7a-7b. 진단 환자(n=794)에서 관찰된 병원균의 분포. 7a. 병원성 하위그룹에 의해 검출된 병원균의 분포; 7b. 균주(환자의 >1% 으로부터 검출된 균주들)에 의해 검출된 병원균의 분포. 분포는 진단된 감염성 질병을 가진 환자들에서 양성 검출의 백분율을 나타낸다.
도 8. 감염성 질병을 가진 것으로 진단된 환자(n=673)에서 관련된 생리학적 시스템의 분포.
도 9a-9b. 임상적 증후군(모든 진단 환자, n=794)의 분포. 9a. 주요 임상적 증후군; 9b. 특이적 임상적 증후군.
도 10. 최대 체온 분포(n=794).
도 11. 증상이 발생한 기간의 분포(n=794). N/A- 데이터를 얻지 못한 건강한 대조군 또는 환자.
도 12a-12b. 환자 집단의 합병증과 관련된 특성. 12a. 합병증의 분포(모든 만성 질환 환자. n=305); 12b. 만성 약물의 분포(모든 만성 질환 환자. n=305). 일부 환자들은 여러 만성 질병을 가지고, 여러 만성 약물로 치료받은 것으로 나타났다.
도 13. 모집군 분포 (진단 환자, n=794)
도 14a-14b. 시그니쳐에 대한 추정 PPV 및 NPV 값 박테리아성 감염 유병률의 함수, 14a. 만장일치 (박테리아성, 바이러스성) 집단 (n=527), 14b. 대다수 (박테리아성, 바이러스성) 집단 (n=653).
도 15a-15b. 대다수 (박테리아성, 바이러스성, 비-감염성) 집단 (n=765)에서 박테리아성, 바이러스성 및 비-감염성 환자들에서의 임상 파라미터 및 실험실 측정의 산탄도. 빨간선 및 원은 각각 그룹 중앙값과 평균에 해당한다. 박테리아성 및 바이러스성 그룹간과 감염성(박테리아성 및 바이러스성) 및 비-감염성(건강한 대상체 포함)간의 T-test p-values를 도시했다.
도 16a-16b. 서로 다른 컷오프를 사용하여 시그니쳐 및 PCT의 성능을 비교. 16a. 만장일치 (박테리아성, 바이러스성) 집단으로부터 76 환자에서 측정된 성능; 16b 대다수 (박테리아성, 바이러스성) 집단으로부터 101 환자에서 측정된 성능. 오류 바는 95% CI를 나타낸다. 시그니쳐 민감도 및 특이성을 한계 면역 반응을 하는 환자의 14%를 필터링한 후 계산하였다.
도 17a-17b. 서로 다른 컷오프를 사용하여 시그니쳐 및 CRP의 성능을 비교. 17a. 만장일치 (박테리아성, 바이러스성) 집단(n=527)으로부터 76 환자에서 측정된 성능; 17b. 대다수 (박테리아성, 바이러스성) 집단 (n=653)로부터 101 환자에서 측정된 성능. 오류 바는 95% CI를 나타낸다. 시그니쳐 민감도 및 특이성을 한계 면역 반응을 하는 환자의 14%를 필터링한 후 계산하였다.
도 18a-18h. 박테리아성 및 바이러스성 환자들에서 선택된 단백질 바이오마커(임의 단위)의 레벨의 산탄도. 빨간선 및 원은 각각 중앙값과 평균에 해당한다. 박테리아성 및 바이러스성 그룹간의 T-test p-values을 도시했다.
도 19a-19b. 시그니쳐의 임상적 정확성은 단백질 측정의 기술적 정확성에서의 감소에 강하다. (19a) 바이러스성 감염으로부터 박테리아성을 구별하는 시그니쳐의 AUC를 TRAIL (y축) 및 CRP (x축) 측정의 CV(std/mean) 함수로써 컬러맵을 사용하여 측정하였다. (19b)도 19A의 대각선 상의 AUC 값은 TRAIL 및 CRP의 CV가 동일하게 표시 되었다.
도 20은 박테리아성 (‘+’), 바이러스성 (‘o’) 및 비-감염성 (‘^’) 환자들의 3차원 시각화이다. 다른 환자들 유형이 CRP (μg/ml), TRAIL 및 IP-10 (pg/ml) 농도 맵에서 구별되는 영역으로 맵핑된다.
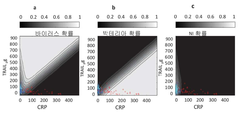
도 21a-21c. 0-100의 IP-10 범위에 대한 본 발명의 일부 실시예에 따라 얻어진 것과 같이, TRAIL (y축), CRP (x축) 및 IP-10 농도의 함수로서 바이러스성(21a), 박테리아성 또는 혼합된(21b) 및 비-감염성 또는 건강한(21c)의 확률.
도 22a-22c. 100-200의 IP-10 범위에 대한 본 발명의 일부 실시예에 따라 얻어진 것과 같이, TRAIL (y축), CRP (x축) 및 IP-10 농도의 함수로서 바이러스성(22a), 박테리아성 또는 혼합된(22b) 및 비-감염성 또는 건강한(22c)의 확률.
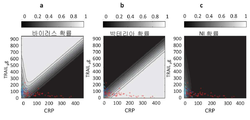
도 23a-23c. 200-300의 IP-10 범위에 대한 본 발명의 일부 실시예에 따라 얻어진 것과 같이, TRAIL (y축), CRP (x축) 및 IP-10 농도의 함수로서 바이러스성(23a), 박테리아성 또는 혼합된(23b) 및 비-감염성 또는 건강한(23c)의 확률.
도 24a-24c. 300-400의 IP-10 범위에 대한 본 발명의 일부 실시예에 따라 얻어진 것과 같이, TRAIL (y축), CRP (x축) 및 IP-10 농도의 함수로서 바이러스성(24a), 박테리아성 또는 혼합된(24b) 및 비-감염성 또는 건강한(24c)의 확률.
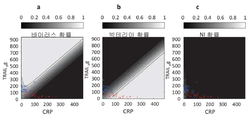
도 25a-25c. 400-500의 IP-10 범위에 대한 본 발명의 일부 실시예에 따라 얻어진 것과 같이, TRAIL (y축), CRP (x축) 및 IP-10 농도의 함수로서 바이러스성(25a), 박테리아성 또는 혼합된(25b) 및 비-감염성 또는 건강한(25c)의 확률.
도 26a-26c. 500-600의 IP-10 범위에 대한 본 발명의 일부 실시예에 따라 얻어진 것과 같이, TRAIL (y축), CRP (x축) 및 IP-10 농도의 함수로서 바이러스성(26a), 박테리아성 또는 혼합된(26b) 및 비-감염성 또는 건강한(26c)의 확률.
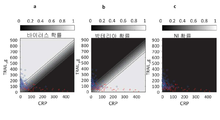
도 27a-27c 1000-2000의 IP-10 범위에 대한 본 발명의 일부 실시예에 따라 얻어진 것과 같이, TRAIL (y축), CRP (x축) 및 IP-10 농도의 함수로서 바이러스성(27a), 박테리아성 또는 혼합된(27b) 및 비-감염성 또는 건강한(27c)의 확률.
도 28a-28c. 2000 이상의 IP-10 범위에 대한 본 발명의 일부 실시예에 따라 얻어진 것과 같이, TRAIL (y축), CRP (x축) 및 IP-10 농도의 함수로서 바이러스성(28a), 박테리아성 또는 혼합된(28b) 및 비-감염성 또는 건강한(28c)의 확률.
도 29a-29f. 본 발명의 일 실시예에 따른 박테리아성 및 비-박테리아성 감염을 구별하는 방법의 예시적인 결과.
도 30a-30b. TRAIL (도 30a) 및 IP-10 (도 30b)의 측정을 위한 빠르고 느린 프로토콜 간의 상관 관계를 나타내는 그래프.
도 31. 본 발명의 다양한 예시적 실시예에 따라 대상체로부터 얻은 생물학적 데이터를 분석하는 적합한 방법의 흐름도.
도 32a-32b. 본 발명의 일부 실시예에 따른 한 평면으로부터 한 표면의 거리를 계산하는 절차를 설명하는 개략도.
도 33a-33d. 본 발명의 일부 실시예에 따른 표면의 한 부드러운 세그먼트를 얻기 위한 절차를 설명하는 개략도.
도 34. 본 발명의 일부 실시예에 따라 생물학적 데이터를 분석하는 시스템의 블록 개략도.
도 35a-35d. 박테리아성 (도 35a), 바이러스성 (도 35b), 비-박테리아성 (도 35c) 및 비-감염성 (도 35d) 병인의 확률을 좌표 δ0 및 δ1의 함수로서 설명하는 등고선 플롯. 상기 확률값 범위는 0% (검은색) to 100% (흰색) 사이이다.
도 36a-36b. 낮은 TRAIL 레벨은 환자의 예후와 결과가 좋지 않고 질병의 심각도가 높다는 표지이다. (36a) 다른 모든 환자(감염성 또는 비-감염성 병인이 있는)와 비교한 ICU에 입원한 환자의 혈청 내 TRAIL 농도. (36b) 감염성 또는 비-감염성 병인이 있는 다른 모든 환자와 비교하여 ICU에 입원했거나 사망한 소아 환자의 혈청 내 TRAIL 농도.
도 37a-37b. 임신 가능한 연령의 남성 및 여성 내 TRAIL 농도 차이를 나타낸 그래프.
도 38a-38e. 본 발명의 일부 실시예에 따라 생물학적 데이터를 분석하기 위한 컴퓨터 구현 방법에서 사용자 입력을 수신하는데 적합한 그래픽 사용자 인터페이스 (GUI)의 스크린 샷.
도 39a-39b. 본 발명의 실시예에서, 네트워크 인터페이스 (도 39a) 및 사용자 인터페이스 (도 39b)를 포함하는 시스템에서 생물학적 데이터를 분석하기 위한 시스템의 블록 개략도.
일부 실시예에서, 본 발명은 컴퓨터 분석법에 관한 것이고, 더 바람직하게는 생물학적 데이터의 컴퓨터 분석법에 관한 것으로, 예를 들어, 박테리아성 감염 및 비-박테리아성 질병간, 및/도는 박테이아성 감염 및 바이러스성 감염간 , 및/또는 감염성 및 비-감염성 질병간을 구별하기 위한 목적의 생물학적 데이터의 분석법에 관한 것이다.
본 발명의 적어도 하나의 실시예에서 상세히 설명하기 전에, 본 발명은 다음의 실시예에 의해 설명되거나 예시된 상세한 적용에 반드시 제한되는 것은 아니라는 점을 알아야 한다. 본 발명은 다른 실시예 또는 다양한 방법으로 실행되거나 수행될 수 있다.
다른 감염성 시료들은 면역 시스템에 의해 인식되거나 목적이 될 수 있는 특이한 분자 패턴을 가진다. 병원성-관련 분자 패턴(PAMPs)은 다른 병원균 그룹과 관련이 있고 Toll-like receptors (TLRs) 및 다른 패턴 인식 수용체 (예, NOD 단백질)를 사용하는 선천성 면역 시스템의 세포에 의해 인식될 수 있는 그러한 분자들의 예시다.
이러한 패턴들은 병원체의 종류에 따라 상당히 다를 수 있고, 이에 따라 다양한 면역 반응을 이끌어낼 수 있다. TLR-4는 그람 음성 박테리아성의 성분인 리포폴리사카라이드(lipopolysaccharide) 및 그람 양성균의 성분인 리포사이코익산(lipoteichoic acids)을 인식하여 면역 시스템의 항균 반응을 촉진시킨다. TLR-3는 (종종 바이러스성 감염을 나타내는) 단일 가닥 RNA를 인식할 수 있으므로 적절한 항 바이러스성 반응을 촉진한다. 서로 다른 분류의 병원체 (예, 박테리아성 대 바이러스성)를 구별함으로써 면역 시스템이 적절한 방어를 할 수 있다.
지난 수십년 동안, 다양한 표지에서 감염원의 감별 진단에 사용될 수 있는 몇몇 숙주 마커가 확인되었다. 병원체가 아닌 숙주에서 추출한 마커를 측정함으로써, 신체의 자연 식물군의 일부인 박테리아성의 비-병원성 균주로 인한 “거짓-긍정(false-positive)” 진단을 최소화할 수 있다. 일 예로 갑상선의 C-세포에서 생성되는 호르몬 칼시토닌의 전구체인 프로칼시토닌(Procalcitonin, PCT)이다. 건강한 개인의 혈류에서 PCT 레벨은 거의 측정되지 않지만 심각한 감염의 결과로 그 레벨이 100 ng/ml까지 급격히 증가할 수 있다. PCT는 전신성 감염, 패혈증을 가진 환자를 진단하는데 많이 사용되고, 민감도가 76 %, 특이도가 70 %이다. 그러나 폐렴이나 상부 호흡기 감염과 같은 다른 비 전신성 감염에서 PCT의 진단적 가치를 테스트한 연구에서 특히 단독으로 사용할 때 제한적이라는 것을 발견했다.
본 발명자들은 이전에 참고 문헌으로 인용된 국제 특허 출원 WO2011132086 및 WO2013/117746에 기재된 바와 같이, 발현 패턴이 감염 유형과 유의하게 관련된 신규 바이오마커 세트를 이전에 확인하였다.
본 발명은, 그 일부 실시예에서, 박테리아성 감염, 바이러스성 감염 및 비-박테리아성, 비-바이러스성 질병의 진단에 폴리펩타이드 시그니쳐의 사용에 기초한다. 본 발명의 실시예의 방법은 적절한 치료법을 선택할 수 있도록, 대상체가 고통받고 있는 감염의 유형을 식별하기 위한 패턴 인식 알고리즘을 사용한다. 본 발명의 다양한 실시예는 다음과 같은 방법으로 현재 진단법의 한계점을 해결한다: (i) 광범위한 병원균에 대한 정확한 진단을 가능; (ii) 신속한 진단 (분 이내) 가능; (ⅲ) 비-병원성 박테리아성 및 바이러스성의 존재에 무감각(거짓-긍정 문제 감소); 및 (iv) 병원체를 직접 채취할 필요성 제거로, 접근하기 어려운 감염을 진단할 수 있다. 따라서, 본 발명의 일부 방법은 항생제 치료가 반드시 필요한 대상체를 선택할 수 있고 단순 바이러스성 감염 또는 비-감염성 질병을 갖는 대상체의 불필요한 항생제 치료를 방지할 수 있다. 본 발명의 일부 방법은 또한 항 바이러스성 치료가 유리한 대상체의 선택이 가능하다.
국제 특허 출원 WO2013/117746에서의 발견을 뒷받침하기 위해, 본 발명자들은 확정된 상이한 유형의 감염을 갖는 1002명의 병원 환자 및 대조군 (비-바이러스성/비- 박테리아성 질병을 가진 환자들 및 건강한 개인)을 등록시켜, 여러 가지 멀티-센터 임상 시험에 참여한 환자의 수를 증가시켰다. 본 발명자들은 이전에 기술된 방법의 정확성 및 민감도를 향상시키기 위해, 환자 (확정된 질병 유형을 가진)를 박테리아성 감염, 바이러스성 감염 및 비-박테리아성, 비-바이러스성 질병의 3 가지 부류 중 하나로 분류하는 3중 분류기(trinary classifier)를 사용하였다. 실시예 3 및 표 9-12에 요약된 바와 같은 2중 분류기(binary classifier)와 비교하였을 때, 본 연구에서 얻은 발현 패턴을 가진 테스트 대상체의 폴리펩타이드의 조합의 레벨이 민감성 및 특이성 측면에서 우수한 결과를 보였다.
본 발명과 관련하여, 다음과 같은 약자가 사용될 수 있다: ANC = 절대 호중구 수(Absolute neutrophil count); ANN = 인공 신경망(Artificial neural networks); AUC = 수신기 작동 곡선 아래의 면적(Area under the receiver operating curve). BP = 보르데텔라 페르투시스(Bordetella pertussis); CHF = 울혈성 심부전(Congestive heart failure); CI = 신뢰 구간(Confidence interval); CID = 선천성 면역 결핍증(Congenital immune deficiency); CLL = 만성 림프성 백혈병(Chronic lymphocytic leukemia); CMV = 사이토메갈로 바이러스성(Cytomegalovirus); CNS = 중추 신경계(Central nervous system); COPD = 만성 폐색성 폐 질환(Chronic obstructive pulmonary disease); CP = 클라미도 필라 폐렴(Chlamydophila pneumonia); CRP = C-반응성 단백질(C-reactive protein); CSF = 뇌척수액(Cerebrospinal fluid); CV = 변동 계수(Coefficient of variation); DOR = 진단 역률(Diagnostic odds ratio); EBV = 엡스타인 바 바이러스성(Epstein bar virus); eCRF = 전자의 경우 보고 양식(Electronic case report form); ED = 응급실(Emergency department), ELISA = 효소 연쇄 면역 흡착 분석(Enzyme-linked immunosorbent assay); FDR = 거짓 발견율(False discovery rate); FMF = 가족성 지중해 열(Familial Mediterranean fever); G-CSF = 과립구 콜로니-자극 인자(Granulocyte colony-stimulating factor); GM-CSF = 과립구-대식세포 콜로니-자극 인자(Granulocyte-macrophage colony-stimulating factor); HBV = B형 간염 바이러스성(Hepatitis B virus); HCV = C형 간염 바이러스성(Hepatitis C virus); HI = 헤모필루스 인플루엔자(Haemophilus influenza); HIV = 인간 면역 결핍 바이러스성(Human immunodeficiency virus); IDE = 전염병 전문가(Infectious disease experts); IL = 인터루킨(Interleukin); IRB = 기관 검토위원회(institutional review board); IVIG = 정맥 면역 글로불린(Intravenous immunoglobulin); KNN = K-근접 이웃(K-nearest neighbors); LP = 레지오넬라 뉴모필라(Legionella pneumophila); LR + = 양의 가능성 비율(Positive likelihood ratio); LR- = 음의 가능성 비율(Negative likelihood ratio); LRTI = 하부 호흡기 감염(Lower respiratory tract infections); mAb = 단클론 항체(Monoclonal antibodies); MDD = 최소 검출 투여량(Minimum detectable dose); MDS = 골수 이형성 증후군(Myelodysplastic syndrome); MP = 마이코플라즈마 뉴모니아(Mycoplasma pneumonia); MPD = 골수 증식성 질환(Myeloproliferative disease); NPV = 음성 예측값(Negative predictive value); PCT = 프로칼시토닌(Procalcitonin); PED = 소아 응급실(Pediatric emergency department); PPV = 양성 예측값(Positive predictive value); QA = 품질 보증(Quality assurance); RSV = 호흡기 세포 융합 바이러스성(Respiratory syncytial virus); RV = 리노바이러스성(Rhinovirus); SIRS = 전신성 염증 증후군(systemic inflammatory syndrome); SP = 스트렙토코커스 뉴모니아(Streptococcus pneumonia); STARD = 진단 정확도의 보고 표준(Standards for Reporting of Diagnostic Accuracy); SVM = 지원 벡터 머신(Support vector machine); TNF = 종양 괴사 인자(Tumor necrosis factor); URTI = 상부 호흡기 감염(Upper respiratory tract infection); UTI = 요로 감염(Urinary tract infection); WBC = 백혈구(White blood cell); WS = 윌콕슨 순위-합계(Wilcoxon rank-sum).
본 발명과 관련하여, 다음과 같은 통계 용어가 사용될 수 있다:
“TP” 는 테스트된 활성을 정확히 반영한 양성 테스트 결과를 의미하는 참 양성이다. 본 발명에서 TP는 박테리아성 감염을 맞게 분류한 것을 포함하고, 이에 제한되지 않는다.
“TN”는 테스트된 활성을 정확히 반영한 음성 테스트 결과를 의미하는 참 음성이다. 본 발명에서 TN은 바이러스성 감염을 맞게 분류한 것을 포함하고, 이에 제한되지 않는다.
“FN”는 음성으로 나타나지만 활성이 명확하지 않은, 거짓 음성이다. 본 발명에서 FN은 실제 박테리아성 감염을 바이러스성 감염으로 잘못 분류한 것의 예시지만, 이에 제한되지 않는다.
“FP”는 양성의 카테고리로 잘못 분류된 테스트 결과를 의미하는 거짓 양성이다. 본 발명에서 FP는 실제 바이러스성 감염을 박테이라성 감염으로 잘못 분류한 것의 예시지만, 이에 제한되지 않는다.
“민감도”는 TP/(TP+FN) 또는 질병 대상체에 대한 참 양성 분율에 의해 계산된다.
“특이성”은 TN/(TN+FP) 또는 비-질병 또는 정상 대상체에 대한 참 음성 분율에 의해 계산된다.
“전체 정확도” 는 (TN + TP)/(TN + FP +TP + FN)에 의해 계산된다.
“양성 예측값” 또는 “PPV”은 TP/(TP+FP) 또는 모든 양성 테스트 결과에 대한 참 양성 분율에 의해 계산된다. 그것은 본질적으로 질병의 유병율 및 테스트 대상 집단에 대한 사전 테스트 확률에 영향을 받는다.
“음성 예측값” 또는 “NPV”는 TN/(TN + FN) 또는 모든 음성 테스트 결과에 대한 참 음성 분율에 의해 계산된다. 그것은 또한 본질적으로 질병의 유병율 및 테스트 대상 집단에 대한 사전 테스트 확률에 영향을 받는다(예, 임상 진단 테스트의 특이성, 민감성 및 양성 및 음성 예측값에 대해 논의한 O’Marcaigh AS, Jacobson RM, "Estimating The Predictive Value Of A Diagnostic Test, How To Prevent Misleading Or Confusing Results,"Clin. Ped. 1993, 32(8): 485-491을 참고).
“MCC(Mathews Correlation coefficient)”는 다음과 같이 계산된다:
MCC = (TP * TN FP * FN) / {(TP + FN) * (TP + FP) * (TN + FP) * (TN + FN)}^0.5
상기 TP, FP, TN, FN는 각각 참-양성, 거짓-양성, 참-음성 및 거짓-음성이다. MCC 값은 각각 완전히 잘못되고 완벽한 분류를 가르키는 -1 내지 +1 사이의 범위를 가진다. MCC가 0인 경우는 랜덤 분류를 의미한다. MCC는 민감도와 특이성을 단일 메트릭에 결합하는 데 유용하다(Baldi, Brunak et al. 2000). 또한 뷴류 크기가 불균형한 경우 분류 정확도를 측정하고 최적화하는데 유용하다(Baldi, Brunak et al. 2000).
"정확도"는 측정 또는 계산된 값(보고된 테스트 값)과 실제 (또는 참) 값의 일치 정도를 나타낸다. 임상적 정확성은 잘못 분류된 결과 (거짓 양성 (FP) 또는 거짓 음성 (FN))와 실제 결과 (참 양성 (TP) 또는 참 음성 (TN))의 비율에 관한 것이고, 여러 측정들 중 민감도, 특이성, 양성 예측값 (PPV) 또는 음성 예측값 (NPV), MCC(Mathews correlation coefficient) 또는 가능성, 오즈 비율(odds ratio), ROC (Receiver Operating Characteristic) 곡선, AUC(Area Under the Curve) 와 같이 기술할 수 있다.
“분석 정확도”는 측정 프로세스 자체의 재현성 및 예측 가능성을 의미하고, 변동 계수 (CV), 피어슨 상관 관계 및 동일한 샘플의 일치성 및 교정에 대한 테스트와 같은 측정들 또는 다른 시간, 사용자, 장비 및/또는 시약 조건들로 요약될 수 있다. 새로운 바이오마커를 평가할 때 고려해야 할 또 다른 고려 사항은 Vasan, 2006에 요약되어 있다.
"성능 (Performance)"은 임상적 및 분석적 정확성, 사용 특성 (예: 안정성, 사용 편의성), 건강의 경제적 가치, 그리고 테스트의 상대적 비용과 같은 기타 분석 및 절차적 특성을 포함하는 진단 또는 예후 테스트의 전반적인 유용성 및 질과 관련된 용어이다. 이러한 요소 중 어느 것이나 우수한 성능의 원천이 될 수 있으며 따라서 테스트의 유용성이 될 수 있으며 AUC 및 MCC, 결과 시간, 유효 기간 등 같은 적합한 "성능 메트릭스"에 의해 측정될 수 있다.
“통계적으로 상당한”의 뜻은 우연히 일어날 것으로 예상되는 것보다 더 큰 변경을 의미한다(“거짓 양성”일 수 있는). 통계적 유의성은 당업계의 공지된 임의의 방법에 의해 결정될 수 있다. 일반적으로 사용되는 유의성의 측정은 데이터 요소가 우연의 결과인 것으로 가정할 때, 주어진 데이터 요소만큼 극단적인 결과를 얻을 확률을 의미하는 p-value를 포함한다. 결과는 종종 0.5 이하의 p-value에서 매우 유의하다고 간주된다.
이하에서 본 발명의 양태를 상세히 설명한다.
도 31은 본 발명의 다양한 예시적 실시예에 따라 대상체로부터 얻은 생물학적 데이터를 분석하는데 적합한 방법의 흐름도이다. 달리 정의하지 않는 한, 이하 기술된 동작들은 많은 조합 또는 실행 순서로 동시에 또는 순차적으로 실행될 수 있다. 특히, 흐름도의 순서는 제한되지 않는다. 예를 들어, 이하 명세서 또는 흐름도에서 특정 순서로 나타나는 하나 이상의 동작들은 다른 순서(예, 역순) 또는 실질적으로 동시에 실행될 수 있다. 추가로, 이하 기술된 일부 동작들은 선택적이고 실행되지 않을 수 있다.
본 발명의 일부 실시예에서 대상체를 미리 항생제로 처리하였고, 본 발명의 일부 실시예에서 대상체를 미리 항생제로 처리하지 않았다.
명세서에 기술된 방법 중 임의의 방법은 많은 형태로 실시될 수 있다. 예를 들어, 상기 방법은 동작들을 수행하기 위해 컴퓨터와 같은 유형의 매체상에서 실시될 수 있다. 상기 방법은 방법 동작들을 수행하기 위해 컴퓨터 판독가능 명령들을 포함하는 컴퓨터 판독 가능 매체상에서 실시될 수 있다. 상기 방법은 컴퓨터 프로그램을 유형의 매체상에서 실행시키거나 컴퓨터 판독 가능 매체상에서 명령을 실행할 수 있도록 배치된 디지털 컴퓨터 성능을 가진 전자 장치상에서 실시될 수 있다.
본 실시예의 방법을 구현하는 컴퓨터 프로그램은 일반적으로 CD-ROM 또는 플래시 메모리 매체와 같은 배포 매체로 사용자들에게 배포될 수 있지만, 이에 제한되는 것은 아니다. 배포 매체로부터 컴퓨터 프로그램은 하드 디스크 또는 유사한 중간 저장 매체에 복사될 수 있다. 본 발명의 일부 실시예에서, 본 발명의 실시예의 방법을 실행하는 컴퓨터 프로그램은 인터넷과 같은 통신 네트워크에 의해 떨어진 위치의 사용자가 상기 프로그램을 다운로드 받음으로써 배포될 수 있다. 컴퓨터 프로그램은 분배 매체 또는 그들의 중간 저장 매체로부터 컴퓨터의 실행 메모리로 컴퓨터 명령을 로딩하여 본 발명의 방법에 따라 작동하도록 컴퓨터를 구성함으로써 실행될 수 있다. 이러한 모든 동작들은 컴퓨터 시스템 분야의 통상의 기술자에게 잘 알려져 있다.
본 발명의 실시예의 방법의 컴퓨터 동작들은 대상체 근처의 서브젝터로부터 멀리 떨어져 있는 컴퓨터에 의해 실행될 수 있다. 컴퓨터가 대상체와 떨어져 있으면 전화 네트워크 또는 인터넷과 같은 네트워크를 통해 데이터를 수신할 수 있다. 이를 위해, 로컬 컴퓨터를 사용하여 멀리 떨어진 컴퓨터에 데이터를 전송할 수 있다. 이러한 구성은 대상체가 다른 위치(예, 집)에 있는 동안 분석이 가능하도록 하고, 다수의 다른 지역에 있는 다수의 대상체에 대한 동시 분석도 가능하게 한다.
상기 방법의 컴퓨터 동작들은 클라우드 컴퓨팅 설비의 클라우드 컴퓨팅 리소스에 의해 실행될 수 있다. 상기 클라우드 컴퓨터 리소스는 컴퓨터 서버 및 선택적으로 저장 서버를 포함할 수 있고, 그리고 당업계에서 잘 알려진 클라우드 컴퓨팅 클라이언트에 의해 운영될 수 있다.
일부 실시예에 따른 상기 방법은 박테리아성 감염을 “확인하는데” 사용될 수 있다. 또한, 상기 방법은 비-박테리아성 감염을 제외하는데 사용될 수 있다. 일부 실시예에 따른 상기 방법은 박테리아성 감염을 “제외하고”, 비-박테리아성 질병을 “확인하는데” 사용될 수 있다.
일부 실시예에 따른 상기 방법은 바이러스성 감염을 “확인하는데” 사용될 수 있다. 또한, 상기 방법은 비-바이러스성 감염을 제외하는데 사용될 수 있다.
일부 실시예에 따른 상기 방법은 바이러스성 감염을 “제외하고” 비-바이러스성 질병을 “확인하는데” 사용될 수 있다.
일부 실시예에 따른 상기 방법은 감염성 질병을 “확인하는데” 사용될 수 있다. 또한, 상기 방법은 비-감염성 질병을 제외하는데 사용될 수 있다. 일부 실시예에 따른 상기 방법은 감염성 질병을 “제외하고” 비-감염성 질병을 “확인하는데” 사용될 수 있다.
상기 방법에 의해 분석된 생물학적 데이터는 대상체의 혈액 중 복수의 폴리펩타이드의 발현값을 포함한다. 일부 실시예에서 생물학적 데이터는 2 개의 폴리펩타이드만의 발현값을 포함하고, 일부 실시예에서 생물학적 데이터는 3 개 이상의 폴리펩타이드의 발현값을 포함하고, 일부 실시예에서 생물학적 데이터는 4개 이상의 폴리펩타이드의 발현값을 포함하고, 일부 실시예에서 생물학적 데이터는 5개 이상의 폴리펩타이드 발현값을 포함하고, 및 일부 실시예에서 생물학적 데이터는 5개의 폴리펩타이드만의 발현값을 포함한다.
본 방명자들은 다양한 유형의 폴리펩타이드를 고려한다. 각각의 예시들은 CRP, IP-10, TRAIL, IL1ra, PCT 및 SAA를 포함하지만 제한되지 않는다. 일부 실시예에서 복수의 폴리펩타이드는 적어도 CRP 및 TRAIL를 포함하고, 및 일부 실시예에서 복수의 폴리펩타이드는 적어도 CRP, TRAIL 및 IP-10를 포함한다.
본 발명의 일부 실시예에서, 생물학적 데이터는 명세서에 추가로 상세히 기술된 바와 같이, 대상체-특이적 데이터 세트의 형태로 제공된다.
특정 실시예에 따라, 분비된(용해된) 폴리펩타이드(예, TRAIL, CRP 및 IP-10)의 레벨은 상기 방법에 의해 분석된다.
본 명세서에서 사용된 용어 “대상체”는 바람직하게는 인간이다. 대상체는 남성 또는 여성일 수 있다. 상기 대상체는 신생아, 유아, 영아 또는 성인일 수 있다. 대상체는 이전에 감염으로 진단받았거나 확인된 인간일 수 있으며, 선택적으로 이미 감염에 대한 치료를 경험했거나 진행 중인 인간일 수 있다. 또한, 대상체는 이미 감염된 것으로 진단받은 인간일 수 있다. 예를 들어, 대상체는 감염에 대한 하나 이상의 위험 인자를 나타내는 인간일 수 있다. 대상체는 감염일수 있으나, 감염의 증상이 없을 수 있다.
본 발명의 일부 실시예에서 따라 진단되는 질병을 가진 대상체는 이하 “테스트 대상체”로 지칭한다. 본 발명자들은 질병이 이미 진단된 복수의 대상체의 폴리펩타이드 발현 패턴에 관한 지식을 수집하고, 수집된 지식에 기초하여 본 발명 실시예의 분석 기술을 고안하였다. 이러한 복수의 대상체는 이하 “이미-진단된 대상체” 또는 “다른 대상체”로 지칭한다.
명세서에 사용된 것과 같이, “박테리아성 감염”은 대상체가 박테리아성로 감염된 상태를 지칭한다. 감염은 증상이 있거나 무증상일 수 있다. 본 발명과 관련하여, 상기 박테리아성 감염은 바이러스성 성분을 포함할 수 있다(박테리아성 및 바이러스성 모두의 결과로 혼합된 감염일 수 있음).
박테리아성 감염은 급성 또는 만성일 수 있다.
급성 감염은 급속한 질병 발병, 상대적으로 짧은 기간의 증상 및 수일 이내에 해결되는 것이 특징이다. 만성 감염은 느리게 진행하고 긴 시간 지속되는 감염이다. 급성 및 만성 감염의 한가지 차이는 급성 감염시 면역 시스템은 감염성 인자에 대응하여 종종 IgM+ 항체를 생산하는 반면에, 만성 감염은 보통 IgM-/IgG+ 항체를 생산한다는 점이다. 추가로, 급성 감염은 면역 매개성 괴사 과정을 일으키지만 만성 감염은 종종 염증 매개성 섬유증 과정 및 징후를 유발한다. 따라서, 급성 및 만성 감염은 다른 근본적인 면역 매커니즘을 유도할 수 있다.
박테리아성 감염은 그람-양성, 그람-음성 박테리아성 또는 비정형 박테리아성에 의해 발생할 수 있다.
상기 용어 “그람-양성 박테리아성”는 펩티도글리칸 및 다당류 및/또는 테이코익산의 세포벽 구조의 일부로서 특성화된 박테리아성를 지칭하며 그람 염색 과정에서의 청자색을 나타내는 것을 특징으로 한다. 대표 그람-양성균은 다음과 같다 : Actinomyces spp ., Bacillus anthracis , Bifidobacterium spp ., Clostridium botulinum , Clostridium perfringens , Clostridium spp ., Clostridium tetani , Corynebacterium diphtheriae , Corynebacterium jeikeium , Enterococcus faecalis , Enterococcus faecium , Erysipelothrix rhusiopathiae , Eubacterium spp ., Gardnerella vaginalis , Gemella morbillorum , Leuconostoc spp ., Mycobacterium abcessus , Mycobacterium avium complex, Mycobacterium chelonae , Mycobacterium fortuitum , Mycobacterium haemophilium , Mycobacterium kansasii , Mycobacterium leprae , Mycobacterium marinum , Mycobacterium scrofulaceum , Mycobacterium smegmatis , Mycobacterium terrae, Mycobacterium tuberculosis, Mycobacterium ulcerans , Nocardia spp ., Peptococcus niger, Peptostreptococcus spp ., Proprionibacterium spp ., Staphylococcus aureus , Staphylococcus auricularis, Staphylococcus capitis , Staphylococcus cohnii , Staphylococcus epidermidis , Staphylococcus haemolyticus , Staphylococcus hominis , Staphylococcus lugdanensis , Staphylococcus saccharolyticus, Staphylococcus saprophyticus , Staphylococcus schleiferi , Staphylococcus similans , Staphylococcus warneri , Staphylococcus xylosus , Streptococcus agalactiae (group B streptococcus), Streptococcus anginosus , Streptococcus bovis , Streptococcus canis , Streptococcus equi , Streptococcus milleri , Streptococcus mitior , Streptococcus mutans , Streptococcus pneumoniae , Streptococcus pyogenes (group A streptococcus), Streptococcus salivarius, Streptococcus sanguis.
상기 용어 “그람-음성 박테리아성”는 각 박테리아성 세포를 둘러싸는 이중막의 존재를 특징으로 하는 박테리아성를 지칭한다.
대표 그람-음성 백테리아는 Acinetobacter calcoaceticus , Actinobacillus actinomycetemcomitans , Aeromonas hydrophila , Alcaligenes xylosoxidans , Bacteroides , Bacteroides fragilis , Bartonella bacilliformis, Bordetella spp ., Borrelia burgdorferi , Branhamella catarrhalis , Brucella spp ., Campylobacter spp ., Chalmydia pneumoniae , Chlamydia psittaci , Chlamydia trachomatis , Chromobacterium violaceum , Citrobacter spp ., Eikenella corrodens , Enterobacter aerogenes , Escherichia coli , Flavobacterium meningosepticum , Fusobacterium spp ., Haemophilus influenzae , Haemophilus spp ., Helicobacter pylori, Klebsiella spp ., Legionella spp ., Leptospira spp ., Moraxella catarrhalis, Morganella morganii , Mycoplasma pneumoniae , Neisseria gonorrhoeae , Neisseria meningitidis, Pasteurella multocida , Plesiomonas shigelloides , Prevotella spp ., Proteus spp ., Providencia rettgeri , Pseudomonas aeruginosa , Pseudomonas spp ., Rickettsia prowazekii , Rickettsia rickettsii, Rochalimaea spp ., Salmonella spp ., Salmonella typhi , Serratia marcescens , Shigella spp., Treponema carateum , Treponema pallidum , Treponema pallidum endemicum , Treponema pertenue , Veillonella spp ., Vibrio cholerae , Vibrio vulnificus , Yersinia enterocolitica 및 Yersinia pestis를 포함한다.
상기 용어 “비정형 박테리아성”는 “그람” 그룹으로 분류되지 못한 박테리아성를 지칭한다. 전형적으로 이들은 세포내 박테리아성 병원체이다. 그들은 Mycoplasmas spp ., Legionella spp . Rickettsiae spp ., and Chlamydiae spp.를 포함하지만, 이에 제한되지 않는다.
상기 용어 “비-박테리아성 질병”은 감염성 박테리아성에 의해 야기된 것이 아닌 질병 또는 상태를 지칭한다.
도 31에 따라, 상기 방법은 310에서 시작하여 제 1 굽은 오브젝트 S의 한 세그먼트 S ROI 및 비-굽은 오브젝트 π 사이의 제 1 거리 d 가 계산되는 311로 진행된다. 일반적으로, 제 1 굽은 오브젝트 S는 n차원에서 매니폴드(manifold)이고, 상기 n은 양의 정수이고, 비-굽은 오브젝트 π는 n+1차원 공간에서 초평면(hyperplane)이다.
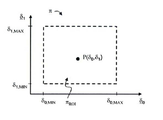
n+1 차원에서 n-차원 매니폴드 및 n+1차원에서 초평면의 개념은 기하학 분야의 통상의 기술자에게 잘 알려져 있다. 예를 들어, n=1 일 때, 제 1 굽은 오브젝트는 곡선이고, 비-굽은 오브젝트 π는 2차원에서 초평면으로, 즉 축을 정의하는 직선이다. n=2일 때, 제 1 굽은 오브젝트는 곡면이고, 비-굽은 오브젝트 π는 3차원 초평면으로, 즉 이하 “평면”으로 지칭되는 편평한 평면이다.
초평면 π는 n방향으로 정의된다. 예를 들어, 상기 비-굽은 오브젝트가 한 축일 때, 단일 방향으로 정의되고, 비-굽은 오브젝트가 평면일 때, 두 방향으로 정의되어, 제 1 방향 및 제 2 방향으로 지칭된다.
매니폴드 S 및 초평면 π 간의 거리는 초평면상의 점 P에서 계산된다. P는 n좌표로 정의된다. 예를 들어, 초평면이 한 축일 때, P는 단일 방향에 따른 단일 좌표 δ1로 정의되고, 그리고 초평면이 평면일 때, P는 (δ0, δ1)로 표시된 좌표 쌍으로 정의되고, 상기 δ0는 “제 1좌표”로 지칭되고 제1 방향에 따라 정의되며, 그리고 δ1는 “제 2 좌표”로 지칭되고 제 2방향에 따라 정의된다. 달리 명시적으로 언급이 없는 한, 좌표 δ0에 대한 참조는 S가 평면이고 π가 평면일 때 고려되는 선택적 실시예를 설명한다.
상기 방향이 이들이 벡터임을 나타내는 밑줄 친 그리스 문자로 표시되는 경우를 제외하고, 상기 방향은 각각의 좌표들과 같이 동일한 그리스 문자로 표시된다. 따라서, 제 1 방향은 δ 0로 표시되고, 상기 제 2 방향은 δ 1로 표시된다.
도 32A는 n=2인 경우에 대한 초평면 π를 나타낸다. 이들 실시예에서, π는 좌표 δ 0 및 δ 1에 의해 정의되는 한 평면이다. 또한 (δ0, δ1)에서 한 점P를 보여준다. 방향 δ 0 및 δ 1는 서로 수직이지만 이것은 반드시 필수적인 것은 아니다. 왜냐하면 상기 δ 0 및 δ 1 사이의 각도는 90과 다를 수 있기 때문이다. 평면 π 안에, δ 0방향을 따라 최소 제 1 좌표 δ0,MIN부터 최대 제 1 좌표 δ0,MAX까지, 및 δ 1방향을 따라 최소 제 2 좌표 δ1,MIN부터 최대 제 2 좌표 δ1,MAX까지 걸친 관심 평면 영역 πROI 이 있다. 점 P는 관심 영역 πROI안에 있다. n=1일 때, π는 한 축이고 관심 영역 πROI은 δ 1 방향에 따른 δ1,MIN 부터 δ1,MAX까지에 걸친 π의 선형 세그먼트이다.
제 1 거리 d의 계산은 초평면 π 및 매니폴드 S를 나타내는 도 32B에 표시된다. 거리 d는 S에서 점 P까지로 π에 수직으로 측정된다. 오브젝트 π 및 S는 각각 1차원의 선으로 도시되어 있지만, π 및 S는 일반적으로 n-차원의 수학적 오브젝트이기 때문에, 이것이 반드시 그럴 필요는 없다는 것을 이해해야 한다. 예를 들어, S가 면이고 π가 평면일 때, π 및 S 모두는 2차원 수학적 오브젝트이다. S의 세그먼트 S ROI 는 관심 영역 πROI 위에 있다. 예를 들어, π가 평면일 때, πROI 는 관심 평면 영역이고, π가 축일 때, πROI 는 축에 따른 선형 세그먼트이다. 따라서, πROI는 π에 대한 SROI 의 투영이다. n=2일 때, SROI은 바람직하게는 (표면)S의 비-평면 세그먼트이고, n=1일 때, SROI 는 바람직하게는 (굽은)S의 굽은 세그먼트이다.
각각의 n 좌표는 폴리펩타이드의 발현값의 조합에 의해 정의된다. 예를 들어, n=1일 때, 좌표 δ1는 폴리펩타이드의 발현값의 조합으로 정의되고, n=2일 때, 좌표 δ0 및 δ1 각각은 폴리펩타이드의 발현값의 상이한 조합에 의해 정의된다.
예를 들어, δ1 및 선택적으로 δ0는 폴리펩타이드의 조합이고, 다음 방정식에 따른다:
δ0 = a 0 + a 1D1 + a 2D2 + ... + φ0
δ1 = b 0 + b 1D1 + b 2D2 + ... + φ1,
여기서, a 0, a 1,... 및 b 0, b 1,...은 일정한 미리 결정된 계수이고, 변수 D1, D2, ... 각각은 폴리펩타이드 중 하나의 발현값이고, φ0 및 φ1은 하나 이상의 발현값에 대한 비선형 함수이다.
함수 φ0 및 φ1각각은 선택적이고, 독립적으로 0으로 설정될 수 있다(또는, 해당 좌표의 계산에 포함되지 않음). φ0=0일 때, 좌표 δ0는 폴리펩타이드의 조합이고, φ1=0일 때, 좌표 δ1는 폴리펩타이드의 조합이다.
비선형 함수 φ0 및 φ1는 선택적이고 바람직하게는 예를 들어, 다음 방정식에 따라 발현값의 멱급수로 표시될 수 있다:
φ0 = Σi q iXi γi
φ1 = Σi r iXi λi,
여기서 i는 합계 지수이고, qi 및 ri는 계수, Xi{D1, D2, ...}들의 세트이며, γi 및 λi 각각은 수치 멱지수이다. 비선형 함수 φ0 및 φ1 각각의 항의 수는 반드시 폴리펩타이드의 수와 같지 않으며, 각각의 합계에서 2 개 이상의 항은 사실 상이한 수치 멱지수를 가짐에도 불구하고 동일한 폴리펩타이드에 상응할 수 있음에 유의한다.
본 실시예에 적합한 계수의 대표적인 예가 다음 실시예 부분에서 제공된다(표 3, 16-20, 32 및 34-39 참고)
φ0=0, φ1=0 및 폴리펩타이드가 TRAIL를 포함하면, δ0는 선택적이고 바람직하게는 TRAIL 발현값의 증가하는 함수이며, 그리고 δ1은 TRAIL의 감소하는 함수이다. φ0=0, φ1=0 및 폴리펩타이드가 CRP를 포함하면, δ1 및 선택적으로 δ0는 선택적으로 바람직하게는 CRP 발현값의 증가하는 함수이다. 폴리펩타이드가 IP-10을 포함하면, δ1 및 선택적으로 δ0는 선택적으로 및 바람직하게는 IP-10 발현값의 증가하는 함수이다.
φ0=0, φ1=0 및 폴리펩타이드가 TRAIL, CRP 및 IP-10를 포함하는 실시예에서, 다음의 방정식에 따라, δ0 및 δ1 각각은 TRAIL, CRP 및 IP-10의 선형 조합일 수 있다:
δ0 = a 0 + a 1C + a 2I + a 3T
δ1 = b 0 + b 1C + b 2I + b 3T,
여기서 C, I 및 T는 각각 CRP, IP-10 및 TRAIL의 발현값이다.
바람직하게는, a1 및 b1 모두 양성이다. 바람직하게는 a2 및 b2 모두 양성이다.
바람직하게는, a3은 양성이고 b3는 음성이다. 조합이 선형 조합이고 폴리펩타이드가 CRP, IP-10 및 TRAIL인 실시예에 적합한 계수의 대표적인 예는 하기 실시예 부분에서 제공된다 (표 3, 16-20 및 36 참고).
φ0=0, φ1=0 및 폴리펩타이드가 TRAIL, CRP 및 IP-10를 포함하는 실시예에서, 다음의 방정식에 따라, δ0 및 δ1 각각은 TRAIL, CRP 및 IP-10의 조합일 수 있다:
δ0 = a 0 + a 1C + a 2I + a 3T + φ0
δ1 = b 0 + b 1C + b 2I + b 3T + φ1,
여기서 φ0 및 φ1 각각은 C, I 및 T 중 적어도 하나 이상의 비선형 함수이다. 대표적인 예로서 φ0 및 φ1은 다음과 같이 표현될 수 있다 :
φ0 = q 1Cγ1 + q 2Cγ2 + q 3Tγ3
φ1 = r 1Cγ1 + r 2Cγ2 + r 3Tγ3.
폴리펩타이드가 CRP, IP-10 및 TRAIL이고 비선형 함수가 0이 아닌 실시예에 대해 적합한 계수의 대표예가 하기 실시예 부분에서 제공된다 (표 39 참고).
πROI의 경계 δ0,MIN, δ0,MAX, δ1,MIN 및 δ1,MAX는 바람직하게는 폴리펩타이드의 발현값의 생리학적으로 가능한 범위에 상응한다.
하기 실시예 8, 더 바람직하게는 실시예 9에 기술된 프로토콜을 사용하여 측정할 때, 생리학적으로 가능한 범위는 전형적으로 0 내지 약 400 ㎍/ml (CRP), 0 내지 약 3000 ㎍/ml (IP-10) 및 0 내지 약 700 pg/ml (TRAIL)이다. 일부 대상체는 이러한 범위를 벗어나는 농도를 나타낼 수 있다. - 본 발명의 다양한 실시예에서, TRAIL, CRP 및 IP-10의 발현값을 하기 실시예 8, 더 바람직하게는 실시예 9에 기술된 프로토콜에 따라 측정할 때, 계수 a 0,..,a 3 및 b 0,...,b 3 은 아래의 표 3에서 취한 것이며, πROI의 경계는 δ0,MIN= -1.3 δ0,MAX=45 δ1,MIN=-14.3 and δ1,MAX=49.6이다.
TRAIL, CRP 및 IP-10의 발현값을 하기 실시예 8, 더 바람직하게는 실시예 9에 기술된 프로토콜과 다른 프로토콜로 측정할 때, 계수 a 0,..,a 3 및 b 0,...,b 3은 아래 표 3의 값과 다르므로 πROI의 경계도 위의 값과 다르다. 이러한 경우, 계수 및 경계의 값은 전술한 값과 상관 관계가 있으며, 여기서 각 계수 및 경계에 대한 상관 관계는 실시예 8, 더 바람직하게는 실시예 9에 기술된 프로토콜에 따라 측정된 각 단백질의 발현값과 실제로 측정된 각 단백질의 발현값간의 관련성으로부터 유래된다.
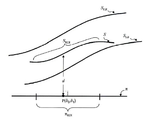
적어도 굽은 오브젝트 S의 세그먼트 SROI 의 주요 부분은 하한 굽은 오브젝트 S LB 및 상한 굽은 오브젝트 S UB으로 지칭되는 2개의 굽은 오브젝트 사이에 있다.
본 명세서에 사용된 "세그먼트 SROI의 주요 부분"은 평활화된 SROI의 길이 (n = 1 일 때), 표면적 (n = 2 일 때) 또는 부피 (n=3 일 때)의 일 부분을 지칭하고 상기 일 부분은 SROI 의 길이, 표면적 또는 부피의 평활화 된 버전의 60 % 또는 70 % 또는 80 % 또는 90 % 또는 95 % 또는 99 % 이다.
본 명세서에서 사용된 "세그먼트 SROI의 평활화된 버전"은 가우스 곡률(Gaussian curvature)이 SROI의 중앙 곡률의 X배인 곡률 임계치를 초과하는 지점 부근의 SROI의 영역을 제외하는 세그먼트 SROI를 지칭하며, 여기서 X는 1.5 또는 2 또는 4 또는 8이다.
다음 절차는 세그먼트 SROI의 주요 부분이 S LB 및 S UB 사이에 있는지 여부를 결정하기 위해 도입될 수 있다. 먼저, 평활화된 세그먼트 SROI 버전을 얻는다. 두번째, 평활화된 세그먼트 SROI 버전의 길이 (n = 1 일 때), 표면적 (n = 2 일 때) 또는 부피 (n3 일 때) A1을 계산한다. 세번째로, S LB 및 S UB 사이에 있는 평활화된 세그먼트 SROI 버전의 일 부분의 길이 (n = 1 일 때), 표면적 (n = 2 일 때) 또는 부피 (n3 일 때) A2를 계산한다. 네번째로, A1에 대한 A2의 백분율을 계산한다.
도 33a-33d는 평활화된 SROI 버전을 얻기 위한 절차를 도시한다.
표현의 명확성을 위해, SROI는 1 차원 세그먼트로서 도시되어 있지만, 통상의 기술자는 SROI가 일반적으로 n 차원의 수학적 오브젝트라는 것을 이해할 것이다. 가우스 곡률은 SROI상의 충분한 수의 표본 점들에 의해 계산된다. 예를 들어 매니폴드가 점 구름(point cloud)으로 표시되면 점 구름의 점에 대해 가우스 곡률을 계산할 수 있다. 그 다음, 가우스 곡률의 중앙값이 얻어지고, 곡률 임계치는 획득된 중앙값에 X 인자를 곱함으로써 계산된다. 도 33A는 평활화되기 이전의 SROI를 도시한다. 가우스 곡률이 곡률 임계치를 초과하는 하나 이상의 지점 (322)을 갖는 영역 (320)이 표시되어 있다. 가우스 곡률이 영역 (320) 내에서 최대인 점 또는 점들이 제거되고, 예를 들어 다항식 보간법(polynomial interpolation)을 통해 영역 (320)이 부드럽게 보간된다 (도 33b). 제거 및 보간은 세그먼트 SROI가 가우스 곡률이 곡률 임계치 이상인 영역을 포함하지 않을 때까지(도 33d) 되풀이하여 반복된다 (도 33c).
n = 1 일 때 (즉 S가 곡선인 경우), S LB는 하한 곡선, S UB는 상한 곡선이다. 이들 실시예에서, S LB 및 S UB는 다음과 같은 형태로 기재될 수 있다 :
SLB = f(δ1)-ε0,
SUB = f(δ1)+ε1
여기서 f(δ1)는 대상체가 박테리아성 감염 가능성을 나타내는 좌표 δ1 (방향 δ 1 을 따른)의 확률 분류 함수이다. 본 발명의 일부 실시예에서, f(δ1)=1/(1+exp(δ1))이다. SLB 및 SUB는 모두 πROI 내의 δ1 값에 대해 양수입니다. 또한, f(δ1)가 테스트 대상체의 바이러스성 감염 가능성을 나타내는 확률 분류 함수인 실시예가 고려된다. 또한, f(δ1)가 테스트 대상체의 감염 가능성을 나타내는 확률 분류 함수인 실시예가 고려된다.
n = 2 일 때 (즉 S가 곡면인 경우), S LB는 하한 곡면, S UB는 상한 곡면이다. 이들 실시예에서, S LB 및 S UB는 다음과 같은 형태로 기재될 수 있다 :
SLB = f(δ0,δ1)-ε0,
SUB = f(δ0,δ1)+ε1
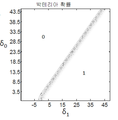
여기서 f(δ0,δ1) 는 대상체가 박테리아성 감염 가능성을 나타내는 (제 1 및 제 2 방향을 따르는) 제 1 및 제 2 좌표의 확률 분류 함수이다. 본 발명의 일부 실시예에서, f(δ0,δ1)=exp(δ1)/(1+exp(δ0)+exp(δ1))이다. S LB 및 S UB는 모두 πROI 내의 δ0 과 δ1의 모든 값에 대해 양수입니다.
상기 실시예들 중 어느 하나에서, 파라미터 ε0 및 ε1 각각은 0.5 미만 또는 0.4 미만 또는 0.3 미만 또는 0.2 미만 또는 0.1 미만 또는 0.05 미만이다.
다시 도 1을 참조하면, 도 31에 도시된 바와 같이, 상기 방법은 상기 계산된 거리 d가 상기 확률 함수 f의 유형에 상응하는 대상체의 질병 또는 상태의 존재 여부 또는 질병 또는 상태의 가능성과 관련되어 있는지 확인하는 312로 진행한다. 예를 들어, 확률 함수 f가 대상체의 박테리아성 감염 가능성을 나타내는 경우, 계산된 거리 d는 대상체의 박테리아성 감염의 존재 여부 또는 박테리아성 감염 가능성과 상관 관계가 있다.
본 발명의 다양한 예시적인 실시예에서, 상관 관계는 거리 d가 대상체의 박테리아성 감염 가능성을 결정함을 포함한다. 가능성은 임의로 그리고 바람직하게는 미리 결정된 임계치 ωB와 비교되고, 상기 방법은 가능성이 ωB 이상일 때 대상체가 박테리아성 감염을 가질 가능성이 있고 대상체가 박테리아성 감염이 없을 것 같지 않음을 결정할 수 있다. ωB의 통상적인 값은 약 0.2, 약 0.3, 약 0.4, 약 0.5, 약 0.6 및 약 0.7를 포함하지만, 이에 한정되는 것은 아니다. 다른 가능성 임계치도 고려된다.
본 발명의 일부 실시예에서, 상기 방법이 대상체에게 박테리아성 감염 가능성이 있다고 결정할 때, 본 명세서에서 추가로 상세히 설명된 바와 같이, 상기 대상체는 박테리아성 감염에 대해 치료된다 (316).
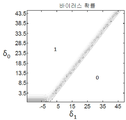
본 발명자들은 테스트 대상체가 바이러스성 감염 가능성을 나타내는 확률 분류 함수 g(δ0,δ1)를 발명했다. 본 발명의 다양한 예시적인 실시예에서, g(δ0,δ1)는 exp(δ0)/(1+exp(δ0)+exp(δ1))와 동일하다.
본 발명의 일부 실시예에 따라, 함수 g는 바이러스성 감염의 존재 여부 또는 바이러스성 감염 가능성을 추정하는데 이용될 수 있다. 따라서, 일부 실시예에서, 상기 방법은 313으로 진행하여 제 2 곡면의 세그먼트 및 평면 π 사이의 제 2 거리를 계산하고, 314에서 제 2 거리는 제 2 곡면의 세그먼트 및 평면 사이의 거리가 대상체의 바이러스성 감염 존재 여부 또는 바이러스성 감염 가능성과 관련있는지 확인한다. 313과 314에 해당하는 절차와 정의는 앞서 설명한 311과 312에 해당하는 절차와 정의와 유사합니다. 따라서, 예를 들어, 제 2 표면의 세그먼트의 주요 부분은 제 2 하한면 g(δ0,δ1)-ε2 및 제 2 상한면 g(δ0,δ1)+ε3 사이에 존재하며, ε2 및 ε3는 각각 0.5 미만 또는 0.4 미만 또는 0.3 미만 또는 0.2 미만 또는 0.1 미만 또는 0.05 미만이다.
본 발명의 일부 실시예에서, 상기 방법은 대상체에게 바이러스성 감염을 가질 가능성이 있다고 결정할 때, 본 명세서에서 추가로 상세히 설명된 바와 같이, 상기 대상체는 바이러스성 감염에 대해 치료된다 (316).
본 발명의 다양한 예시적인 실시예에서, 상관 관계는 제 2거리가 대상체의 바이러스성 감염 가능성을 결정함을 포함한다. 가능성은 임의로 그리고 바람직하게는 미리 결정된 임계치 ωV와 비교되고, 상기 방법은 가능성이 ωV 이상일 때 대상체가 바이러스성 감염을 가질 가능성이 있고 대상체가 바이러스성 감염이 없을 것 같지 않음을 결정할 수 있다. ωV의 통상적인 값은 약 0.5, 약 0.6, 약 0.7 및 약 0.8를 포함하지만, 이에 한정되는 것은 아니다. 다른 가능성 임계치도 고려된다.
313 및 314 과정이 실행되는 실시예들에서, 311 및 312 과정은 실행되거나 실행되지 않을 수 있다. 예를 들어, 본 실시예는 311 및 312 과정이 실행되지 않는 절차를 고려하고, 상기 방법은 제 1 거리를 계산하지 않고 및 제 1 거리와 대상체의 박테리아성 감염 존재 유무 또는 박테리아성 감염 가능성과의 상관관계와 관련없이, 대상체의 바이러스성 감염 가능성을 결정한다.
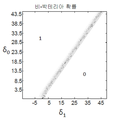
또한, 모든 311 내지 314 과정 실행될 수 있으며, 여기서 311 및 312는 314의 결과와 무관하게 실행되고, 313 및 314는 312의 결과와 무관하게 실행된다. 이러한 실시예들에서, 상기 방법은 선택적으로 및 바람직하게는 대상체의 박테리아성 감염 가능성 및 바이러스성 감염 가능성을 각각 결정한다. 이들 가능성 각각은 미리 결정된 임계치 (ωB 또는 ωV)와 비교될 수 있다. 가능성의 각각이 각각의 역치값보다 낮으면, 이 방법은 대상체가 비 박테리아성 및 비 바이러스성 감염성 질병을 가질 가능성이 있다고 결정할 수 있다. 예를 들어, 상기 방법은 대상체가 비 감염성 질병, 진균성 질병 또는 기생충 질병을 가질 가능성이 있음을 결정할 수 있다.
또한, 일부 과정들이 실행되는지의 여부는 하나 이상의 다른 과정들의 결과에 의존한다. 예를 들어, 상기 방법은 대상체의 박테리아성 감염 가능성을 결정하기 위해 311 및 312를 실행할 수 있다. 그 후, 결정된 가능성은 임계값 ωB와 비교된다. 상기 방법은 결정된 가능성이 ωB를 초과하면 313 및 314의 실행을 스킵하고, 그렇지 않으면 313 및 314를 실행한다. 이들 실시 형태의 다른 예는 상기 방법이 313 및 314를 실행하여 대상체의 바이러스성 감염 가능성을 결정하는 절차이다. 그 후, 결정된 가능성은 임계값 ωV와 비교된다. 상기 방법은 결정된 가능성이 ωV를 초과하면 311 및 312의 실행을 스킵하고, 그렇지 않으면 311 및 312를 실행한다.
상기 방법은 가능성의 결과가 생성되는 315 단계로 선택적으로 및 바람직하게는 계속된다. 결과는 문자 및/또는 그래픽 및/또는 컬러 인덱스을 사용하여 표시할 수 있다. 결과에는 선택적으로 임계값 ωB에 대한 비교 결과가 포함될 수 있다. 도 29a-29f 및 도 38a-38e는 본 발명의 일 실시예에 따른 박테리아성 및 비 박테리아성 감염을 구별하는데 적합한 예시적인 결과를 도시한다.
상기 방법은 317에서 끝난다.
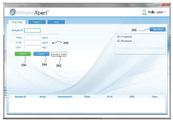
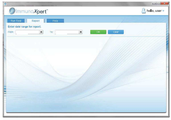
도 38a-38e는 본 발명의 일부 실시예에 따라 생물학적 데이터를 분석하기 위한 컴퓨터 구현 방법에서 사용자 입력을 수신하기에 적합한 그래픽 사용자 인터페이스 (GUI)의 스크린 샷이다.
GUI는 버튼 제어의 형태일 수 있는 계산 활성 제어부 (390)를 포함한다. GUI는 또한 복수의 발현값 입력 필드 (380)를 포함할 수 있으며, 각 발현값 입력 필드는 사용자로부터 대상체의 혈액 내의 폴리펩타이드의 발현값을 수신하도록 구성된다. 사용자는 입력란에 폴리펩타이드의 발현값을 입력한다. 또한, 발현값은 컴퓨터 및 발현값을 측정하는 외부 기계 (도시되지 않음) 사이의 통신을 수립함으로써 수신될 수 있다. 이들 실시예에서, 사용자가 입력 필드에 발현값을 수동으로 입력할 필요는 없다. 일부 실시예에서, GUI는 예컨대, 버튼 제어의 형태인 통신 제어부 (392)를 포함하며, 여기서 외부 기계와의 통신은 사용자에 의한 통신 제어의 활성에 대한 응답이다.
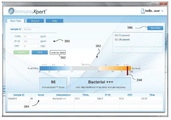
사용자에 의한 제어 (390)의 활성에 응답하여, 컴퓨터는 자동적으로 또는 필드 (380)를 통해 수신된 발현값에 기초하여 점수를 계산한다. 상기 점수는 대상체가 박테리아성 감염 및/또는 바이러스성 감염을 가질 가능성일 수 있다. 점수는, 예를 들어 상술한 바와 같이 곡면과 두 방향에 의해 정의된 평면 사이의 거리를 계산함으로써 계산될 수 있다.
그래픽 스케일 (382)은 GUI 상에 생성될 수 있다. 그래픽 스케일은 바이러스성 감염에 대응하는 것으로 식별된 제 1 말단, 및 박테리아성 감염에 대응하는 것으로 식별된 제 2 말단을 포함할 수 있다.
일단 점수가 계산되면, 임의로 그리고 바람직하게는 계산된 가능성에 대응하는 위치에서 그래픽 (382) 상에 마크 (394)가 만들어 질 수 있다. 도 38a는 입력 필드에 값들이 입력되기 전의 GUI를 나타낸다. 도 38B는 박테리아성 감염일 96 %의 가능성에 상응하는 위치에서 스케일 (382)상의 마크 (394)를 나타낸다. 도 38c는 박테리아성 감염 (또는 균등하게 감염이 바이러스성 성일 확률)일 1 %의 가능성에 해당하는 위치에서 스케일 (382)상의 마크 (394)를 나타낸다. 선택적으로 상기 GUI는 계산된 점수를 숫자로 표시한다.
GUI는 임의로 및 바람직하게 버튼 제어의 형태일 수 있는 하나 이상의 추가 제어부 (386, 388)를 포함한다. 예를 들어, 제어부 (388)는 사용자가 제어부 (388)를 활성화할 때 입력 필드 (380)의 내용을 지우도록 컴퓨터에 명령할 수 있다. 이는 사용자가 다른 샘플에 대응하는 값을 입력하게 한다. 일부 실시예에서, GUI는 또한 이전 샘플의 결과를 요약하는 결과 (384)를 생성한다. 제어부 (386)는 사용자가 제어부 (386)를 활성화 할 때 입력 필드 (380)뿐만 아니라 결과 (384)를 지우도록 컴퓨터에 명령할 수 있다. 이는 사용자가 GUI로부터 로그 아웃하지 않고 새로운 실행을 시작할 수 있게 한다.
본 실시예에 적합한 프로토콜의 대표예는 다음과 같다.
GUI는 사용자가 실험의 품질 관리 (QC) 값을 입력할 수 있는 대화 상자를 인증된 사용자에게 제공한다. QC가 검증되고, GUI가 도 38A에 생성된다. 사용자는 필드 (380)에 발현값을 입력하고 제어부 (390)를 활성화하여 결과 (예를 들어, 도 38b 및 38c)를 수신한다. 다른 혈액 샘플의 표현 값을 공급하기 위해, 사용자는 컨트롤 (388)을 활성화시킨다. 각 샘플의 결과는 예를 들어 테이블의 형태일 수 있는 결과 (384)에 추가된다. 소프트웨어를 닫지 않고 새 실험에 들어가거나 사용자가 로그 아웃하려면 제어부 386을 활성화하여 결과 384를 지우고 새 QC 값을 입력해야 한다. 바람직하게는, 모든 작업이 하나 이상의 로그 파일에 기록된다.
본 발명의 일부 실시예에서, GUI는 또한 예를 들어 날짜 기반 요청에 응답하여 이전 실험의 결과를 표시하는 보고서 화면 (도 38d 및 38e)을 포함한다.
본 명세서에 제시된 폴리펩타이드 명칭이 예로서 주어진 것으로 이해될 것이다. 많은 다른 명칭, 별칭, 변형, 이성질체 및 변형이 통상의 기술자에게 명백할 것이다. 따라서 모든 대체 단백질 이름, 별칭, 변형 이성체 및 변형을 포함하는 것으로 간주된다.
유전자 생성물은 국제 인간 게놈 기구 명명위원회(HGNC)가 지정한 공식 문자 약어 또는 유전자 기호에 따라 식별되며 Entrez Gene로 잘 알려져 있는 미국 국립 생명 공학 정보 센터 (NCBI) 웹 사이트에서 제출한 날짜에 기재되어 있다.
TRAIL : 이 유전자에 의해 암호화된 TNF 관련 세포 사멸 유발 리간드 (TNF Related Apoptosis Inducing Ligand, TRAIL) 단백질은 종양 괴사 인자 (TNF) 리간드 계열에 속하는 사이토카인이다. 유전자의 다른 이름은 APO2L, TNF- 관련 세포 사멸-유발 리간드, TNFSF10 및 CD253을 제한없이 포함한다. TRAIL은 멤브레인 결합 형태 및 가용성 형태로 존재하며, 이들 모두는 형질 전환된 종양 세포와 같은 상이한 세포에서 세포사멸를 유도할 수 있다. 이 단백질은 TNFRSF10A/TRAILR1, NFRSF10B/TRAILR2, NFRSF10C/TRAILR3, TNFRSF10D/TRAILR4 및 가능하면 NFRSF11B/OPG와 같은 TNF 수용체 슈퍼 패밀리의 일부 구성원에 결합한다. 이 단백질의 활성은 세포 사멸을 유도할 수 없는 NFRSF10C/TRAILR3, TNFRSF10D/TRAILR4 및 NFRSF11B/OPG와 같은 디코이(decoy) 수용체에 결합함으로써 조절될 수 있다. MAPK8/JNK, caspase 8 및 caspase 3의 활성화를 유도하기 위해 이 단백질의 수용체에 결합하는 것으로 보여진다. 다른 이성체를 암호화하는 다른 스플라이싱된 전사 변이가 이 유전자에서 발견되었다. TRAIL은 균질 구조를 갖는 가용성 형태를 생성하기 위해 세포 표면으로부터 분해에 의해 절단될 수 있다.
특정 실시예에 따라, TRAIL의 가용성(즉, 분비된) 형태의 값이 측정된다.
다른 실시예에 따라, TRAIL의 멤브레인 형태가 측정된다.
또 다른 실시예에 따라, TRAIL의 멤브레인 형태 및 TRAIL의 분비 된 형태 모두가 측정된다.
본 발명의 또 다른 측면에 따라, 감염 유형을 나타내는 가용성 TRAIL 및 불용성 TRAIL의 농도를 측정하는 단계를 포함하는 대상체의 감염 유형을 결정하는 방법을 제공한다.
일 실시예에서, 가용성 TRAIL의 농도가 미리 결정된 역치값보다 높을 때, 박테리아성 감염은 상기 대상체에 대해 배제된다.
또 다른 실시예에서, 가용성 TRAIL의 농도가 미리 결정된 역치값보다 높을 때, 바이러스성 감염이 대상체에 대해 결정된다.
가용성 TRAIL에 대한 예시적인 단백질 서열은 서열번호 37 및 서열번호 38로 표시된다.
멤브레인 인간 TRAIL의 예시적인 mRNA 서열은 서열번호 1로 표시된다.
멤브레인 인간 TRAIL의 예시적인 아미노산 서열은 서열번호 4로 표시된다.
TRAIL에 대한 다른 예시적인 cDNA 및 아미노산 서열은 서열번호 2, 3 및 5-8로 표시된다.
IP10 : 이 유전자는 CXC 서브 패밀리의 케모카인 및 수용체 CXCR3에 대한 리간드를 암화화한다. 이 단백질을 CXCR3에 결합시키면 단핵 세포의 자극(stimulation of monocytes), 자연 살해자(natural killer) 및 T 세포 이동(T-cell migration), 접착 분자 발현 조절(modulation of adhesion molecule expression) 등의 다면 발달 효과가 나타난다. 유전자의 다른 이름은 IP-10, CXCL10, 감마-IP10, INP10 및 케모카인 (C-X-C 모티브) 리간드 10을 포함하고, 이에 제한되지 않는다..
인간 IP10의 예시적인 cDNA 서열은 서열번호 9-12로 표시된다. 인간 IP10의 예시적인 아미노산 서열은 서열번호 13으로 표시된다.
CRP : C-반응성 단백질; CRP의 다른 별칭에는 RP11-419N10.4 및 PTX1이 포함된다. 이 유전자에 의해 암호화되는 단백질은 펜타신 패밀리에 속한다. 그것은 외부 병원체 및 숙주의 손상된 세포를 인식하고 혈액 내의 체액성 및 세포성 이펙터 시스템과 상호 작용하여 그들의 제거를 시작하는 능력에 기초하여 여러 가지 숙주 방어 관련 기능에 관여합니다. 따라서 조직 손상, 감염 또는 기타 염증성 자극에 대한 급성기 반응에서 혈장 내 단백질의 레벨이 크게 증가합니다. CRP는 숙주 방어와 관련된 몇 가지 기능을 다음과 같이 나타낸다 : 그것은 포스포릴콜린에 대한 칼슘 의존성 결합을 통해 응집, 박테리아성의 캡슐 팽창, 식균 작용 및 보체 고정을 촉진한다.
인간 CRP의 예시적인 cDNA 서열은 서열번호 14-16로 표시된다.
인간 CRP의 예시적인 아미노산 서열은 서열번호 17로 표시된다.
IL1RA : 이 유전자에 의해 암호화되는 단백질은 인터루킨 1 수용체 패밀리에 속하는 사이토카인 수용체이다. 이 단백질은 인터루킨 알파 (IL1A), 인터루킨 베타 (IL1B) 및 인터루킨 1 수용체, 타입 I (IL1R1 / IL1RA)의 수용체이다. 이것은 많은 사이토카인 유도 면역 및 염증 반응에 관여하는 중요한 중재자이다. 유전자의 추가적인 이름은 CD121A, IL-1RT1, p80, CD121a 항원, CD121A, IL1R 및 IL1ra를 포함하고, 이에 제한되지 않는다.
인간 IL1RA의 예시적인 cDNA 서열은 서열번호 18, 19 및 20로 표시된다.
인간 IL1RA의 예시적인 아미노산 서열은 서열번호 21 내지 24로 표시된다.
PCT : 프로칼시토닌 (Procalcitonin, PCT)은 호르몬 칼시토닌의 펩타이드 전구체이며 후자는 칼슘 항상성과 관련이 있다. 프로칼시토닌 (Procalcitonin, "pCT")은 116 개의 아미노산으로 구성되고 약 13,000 달톤의 분자량을 갖는 단백질이다. 그것은 갑상선의 C 세포에 의해 정상적인 대사 조건 하에서 생성되고 분비되는 칼시토닌의 프로호르몬이다. pCT 및 칼시토닌 합성은 141 개의 아미노산을 포함하는 전구체 펩타이드인 프리-칼시토닌 (pre-pCT)의 번역에 의해 개시된다. 인간 pre-pCT의 아미노산 서열은 1984 년 Moullec et al. FEB Letters, 167 : 93-97에 게재되었다. pCT는 신호 펩타이드 (pre-pCT의 처음 25 개 아미노산)의 절단 후에 형성된다.
인간 PCT의 예시적인 cDNA 서열은 서열번호 31-32로 표시된다.
인간 PCT의 예시적인 아미노산 서열은 서열번호 33-36으로 표시된다.
SAA : 아포리포단백질(apolipoproteins)의 혈청 아밀로이드 A 패밀리의 구성원을 암호화한다. 암호화된 단백질은 염증 및 조직 손상에 대한 반응으로 고도로 발현되는 주요 급성기 단백질이다. 이 단백질은 또한 HDL 대사 및 콜레스테롤 항상성에 중요한 역할을 한다. 이 단백질의 높은 수치는 동맥 경화증, 류마티스 관절염, 알츠하이머병 및 크론 병과 같은 만성 염증성 질환과 관련이 있다. 이 단백질은 특정 종양의 잠재적인 바이오 마커 일 수도 있다. 교대로 스플라이싱하면 동일한 단백질을 암호화하는 여러 가지 전사 변이가 생긴다.
인간 SAA의 예시적인 cDNA 서열은 서열번호 25 내지 27로 표시된다.
인간 SAA의 예시적인 아미노산 서열은 서열번호 28-30으로 표시된다.
환자 대 환자의 DNA 변이가 단백질의 아미노산 서열의 차이를 야기 할 수 있는 SNP를 야기할 수 있기 때문에, 본 발명자는 또한 아미노산 서열이 상기 제공된 서열에 적어도 90 %, 95 % 또는 99 % 상동성을 가지는 단백질을 고려하였다.
폴리펩타이드 (예를 들어, TRAIL, IP-10 및 CRP) 레벨을 측정하는 것은 전형적으로 하기에 기술되는 바와 같이 단백질 수준에서 영향을 받는다.
단백질들의 발현 및/또는 활성을 검출하는 방법
본 발명의 일부 실시예의 배양 세포에서 발현된 단백질의 발현 및/또는 활성 레벨은 당업계에 공지된 방법을 사용하여 결정할 수 있으며, 전형적으로는 항체의 사용을 포함할 수 있다. 이러한 방법은 면역 분석법 (immunoassays)이라고 할 수 있다. 면역 분석법은 시약을 첨가하고 세척하거나 분석법 내에서 다른 지점에서 분리하여 여러 단계로 실행할 수 있다. 다중 단계 분석법은 종종 분리 면역 분석법 또는 이종 면역 분석법으로 불린다. 일부 면역 분석법은 단순히 시약과 시료를 혼합하고 물리적 측정을 수행함으로써 수행될 수 있다. 이러한 분석법을 균질 면역 분석법 또는 비분리 면역 분석법이라고 한다. 교정기(Calibrators)의 사용은 종종 면역 분석법에 사용됩니다. 교정기는 해당 분석물을 포함하고 있는 것으로 알려진 용액이며 일반적으로 분석물의 농도는 알려져 있다. 교정기에 의해 생성된 분석의 반응물에 대한 실제 샘플에 대한 분석의 반응물 간의 비교는 샘플 내 분석물의 존재 또는 농도와 관련하여 신호 강도를 해석하는 것을 가능하게 한다.
항체는 단클론성, 다클론성, 키메라성 또는 이들의 단편일 수 있으며, 반응물을 검출하는 단계는 임의의 적합한 면역 분석법으로 수행 될 수 있다.
폴리펩타이드의 검출을 위한 항원에 대한 적합한 소스는 상업적으로 이용 가능한 소스를 포함하고, 예를 들어 Abazyme, Abnova, AssayPro, Affinity Biologicals, AntibodyShop, Aviva bioscience, Biogenesis, Biosense Laboratories, Calbiochem, Cell Sciences, Chemicon International, Chemokine, Clontech, Cytolab, DAKO, Diagnostic BioSystems, eBioscience, Endocrine Technologies, Enzo Biochem, Eurogentec, Fusion Antibodies, Genesis Biotech, GloboZymes, Haematologic Technologies, Immunodetect, Immunodiagnostik, Immunometrics, Immunostar, Immunovision, Biogenex, Invitrogen, Jackson ImmunoResearch Laboratory, KMI Diagnostics, Koma Biotech, LabFrontier Life Science Institute, Lee Laboratories, Lifescreen, Maine Biotechnology Services, Mediclone, MicroPharm Ltd., ModiQuest, Molecular Innovations, Molecular Probes, Neoclone, Neuromics, New England Biolabs, Novocastra, Novus Biologicals, Oncogene Research Products, Orbigen, Oxford Biotechnology, Panvera, PerkinElmer Life Sciences, Pharmingen, Phoenix Pharmaceuticals, Pierce Chemical Company, Polymun Scientific, Polysiences, Inc., Promega Corporation, Proteogenix, Protos Immunoresearch, QED Biosciences, Inc., R&D Systems, Repligen, Research Diagnostics, Roboscreen, Santa Cruz Biotechnology, Seikagaku America, Serological Corporation, Serotec, SigmaAldrich, StemCell Technologies, Synaptic Systems GmbH, Technopharm, Terra Nova Biotechnology, TiterMax, Trillium Diagnostics, Upstate Biotechnology, US Biological, Vector Laboratories, Wako Pure Chemical Industries, 및 Zeptometrix이 있다. 그러나, 통상의 기술자는 본 명세서에 기재된 임의의 폴리펩타이드에 대한 항체를 일상적으로 제조할 수 있다.
폴리펩타이드를 측정하기 위한 다클론 항체는 토끼, 염소, 양, 닭, 오리, 기니아 돼지, 마우스, 당나귀, 낙타, 랫트 및 말 중 하나 이상의 능동 면역에 의해 혈청으로부터 생산된 항체를 포함하고, 이에 제한되지 않는다.
추가적인 검출 시약의 예로는 scFv, dsFv, Fab, sVH, F(ab')2, Cyclic peptides, Haptamers, A single-domain antibody, Fab fragments, Single-chain variable fragments, Affibody molecules, Affilins, Nanofitins, Anticalins, Avimers, DARPins, Kunitz domains, Fynomers 및 Monobody을 포함하고, 이에 제한되지 않는다.
Enzyme linked immunosorbent assay (ELISA): ELISA를 수행하는 것은 특정 항원에 대해 특이성을 갖는 적어도 하나의 항체를 포함한다. 미지의 항원을 가진 샘플을 비-특이적으로 (표면 흡착을 통해) 또는 특이적으로 (동일한 항원에 특이적인 다른 항체에 의한 포획을 통해, "샌드위치 (sandwich)"형태로) 고체 지지체 (일반적으로 폴리스티렌 마이크로 타이터 플레이트) "ELISA)에 고정시킨다. 항원이 고정화된 후, 검출 항체가 첨가되어 항원과 복합체를 형성한다. 검출 항체는 효소에 공유 결합 될 수 있거나, 생체 접합을 통해 효소에 연결된 2 차 항체에 의해 검출될 수 있다. 각 단계 사이에서, 플레이트는 일반적으로 약한 세제 용액으로 세척되어 비특이적으로 결합된 모든 단백질 또는 항체를 제거한다. 최종 세척 단계 후, 플레이트는 효소 기질을 첨가하여 샘플 내의 항원 양을 나타내는 가시적 신호를 생성함으로써 전개된다.
이 방법에서 일반적으로 사용되는 효소는 양 고추냉이 퍼옥시다제 및 알칼라인 포스파타제를 사용한다. 잘 교정되고 선형 응답 범위 내에서 샘플에 존재하는 기질의 양은 생성된 색의 양에 비례한다. 기질 표준은 일반적으로 정량적 정확도를 향상시키기 위해 사용된다.
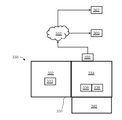
Western blot: 이 방법은 아크릴아마이드겔에 의해 다른 단백질로부터 기질을 분리한 후 기질을 멤브레인 (예 : 나일론 또는 PVDF)으로 옮기는 것을 포함한다. 이어서, 기질의 존재는 기질에 특이적인 항체에 의해 검출되고, 이어서 항체 결합 시약에 의해 검출된다. 항체 결합 시약은 예를 들어 단백질 A 또는 다른 항체일 수 있다. 항체 결합 시약은 전술한 바와 같이 방사성으로 표지되거나 효소가 연결될 수 있다. 검출은 오토라디오그래피(autoradiography), 비색 반응(colorimetric reaction) 또는 화학 발광(chemiluminescence)에 의한 것 일 수 있다. 이 방법은 전기 영동 동안 아크릴아미드겔에서 이동 거리를 나타내는 멤브레인 상의 상대 위치에 의한 기질의 양의 정량 및 그의 동일성의 결정을 허용한다.
Fluorescence activated cell sorting ( FACS ): 이 방법은 기질 특이적 항체에 의해 세포 내에서 기질을 검출하는 것을 포함한다. 기질 특이적 항체는 형광 표지에 연결된다. 탐지는 빛을 통과할 때 각 세포에서 방출되는 빛의 파장을 읽는 세포 선별기를 사용한다. 이 방법은 2 개 이상의 항체를 동시에 사용할 수 있다.
Automated Immunoassay: 면역분석법 (종종 "Automated Immunoassay"라고도 함)에 적용된 자동화 된 분석기는 인간의 도움을 최소화하면서 다양한 생물학적 샘플에서 다양한 화학 물질 및 기타 특성을 신속하게 측정하도록 설계된 의료 실험 장비입니다. 측정된 이러한 혈액 및 기타 체액의 특성은 질병의 진단에 유용 할 수 있다. 분석기에 샘플을 도입하는 많은 방법이 발명되었다. 이것은 샘플의 시험관을 궤도를 따라 움직일 수 있는 랙에 놓거나 샘플을 사용 가능하게 만들기 위해 회전하는 원형 회전 장치에 튜브를 삽입하는 것과 관련될 수 있다. 일부 분석기는 샘플을 샘플 컵에 옮겨야 한다. 그러나 실험실 직원의 건강과 안전을 보호하려는 노력으로 많은 제조업체가 밀폐된 튜브 샘플링 기능을 갖춘 분석기를 개발하여 작업자가 샘플에 직접 노출되는 것을 방지했다. 샘플은 개별적으로, 일괄적으로 또는 연속적으로 처리할 수 있다.
자동화된 면역분석법 기계의 예는 다음을 포함하고, 이에 제한되지 않는다: ARCHITECT ci4100, ci8200 (2003), ci16200 (2007), ARCHITECT i1000SR, ARCHITECT i2000,, i2000SR, i4000SR,
AxSYM/AxSYM Plus, 1994 U.S.,
DS2,
AIMS,
AtheNA, DSX, ChemWell, UniCel DxI 860i Synchron Access Clinical System, UniCel DxC 680i Synchron Access Clinical System, Access/Access 2 Immunoassay System, UniCel DxI 600 Access Immunoassay System, UniCel DxC 600i Synchron Access Clinical System, UniCel DxI 800 Access Immunoassay System, UniCel DxC 880i Synchron Access Clinical System, UniCel DxI 660i Synchron Access Clinical System, SPA PLUS (Specialist Protein Analyzer), VIDAS Immunoassay Analyzer, BioPlex 2200, PhD System EVOLIS PR 3100TSC Photometer, MAGO 4S/2011 MagoPlus Automated EIA Processor, LIAISON XL/2010 LIAISON, ETI-MAX 3000 Agility, Triturus, HYTEC 288 PLUSDSX, VITROSECi Immunodiagnostic System, VITROS 3600 Immunodiagnostic System, Phadia Laboratory System 100E, Phadia Laboratory System 250, Phadia Laboratory System 1000, Phadia Laboratory System 2500, Phadia Laboratory System 5000, cobas e 602/2010, cobas e411, cobas e601, MODULAR ANALYTICS E170, Elecsys 2010, Dimension EXL 200/2011, Dimension Xpand Plus Integrated Chemistry System, Dimension RxL Max/Max SuiteIntegrated Chemistry System,; Dimension RxL Integrated Chemistry System, Dimension EXL with LM Integrated Chemistry System, Stratus CSAcute Care Diagnostic System, IMMULITE 2000 XPi Immunoassay System, ADVIA Centaur CP Immunoassay System, IMMULITE 2000, IMMULITE 1000, Dimension Vista 500 Intelligent Lab System,
Dimension Vista 1500 Intelligent Lab System, ADVIA Centaur XP,
AIA-900, AIA-360, AIA-2000, AIA-600 II, AIA-1800.
CRP, IP-10 및 TRAIL의 측정은 Luminex machine상에서도 수행될 수 있다.
레터럴 플로우 면역분석법(Lateral Flow Immunoassays, LFIA ): 이것은 POC (point of care)에서 분석물의 신속한 측정을 가능하게 하는 기술이며, 그 기본 원리는 이하 설명되어 있다. 일 실시예에 따르면, LFIA는 휴대용 장치와 관련하여 사용된다.
이 기술은 다공성 종이 또는 소결 폴리머와 같은 일련의 모세혈관계을 기반으로 한다. 이들 요소 각각은 유체 (예: 소변)를 자발적으로 운반할 수 있는 능력을 가지고 있다. 첫 번째 요소 (샘플 패드)는 스폰지 역할을 하여 과량의 샘플 유체를 보유한다. 일단 담기면 유체는 두 번째 요소 (컨쥬게이트 패드)로 옮겨져 제조자가 소위 컨쥬게이트 (conjugate) 즉, 표적 분자(예: 항원) 및 입자 표면에 고정된 화학적 파트너(예 : 항체) 간의 최적화된 화학 반응을 보장하는 모든 것을 포함하는 염-당 매트릭스에 건조된 형태의 생체 활성 입자(아래 참조)를 저장한다. 샘플 유체가 염-당 매트릭스를 용해시키는 동안, 그것은 또한 입자를 용해시키고 하나의 결합된 수송에서 샘플 및 컨쥬게이트는 다공성 구조를 통해 유동하면서 혼합된다. 이러한 방식으로, 분석물은 제 3 모세혈관계을 통해 더 이동하면서 입자들과 결합한다. 이 물질은 제조자에 의해 고정된 제 3 분자의 하나 이상의 영역 (종종 스트립이라고도 함)을 갖는다. 샘플-컨쥬게이트 혼합물이 이들 스트립에 도달할 때까지, 분석물은 입자와 결합하고, 제 3 '포획 (capture)' 분자는 복합체와 결합한다.
잠시 후, 점점 많은 입자들이 스트립을 통과하면, 입자가 쌓이고 스트립 영역의 색깔이 변한다. 일반적으로 2개 이상의 스트립이 있다 : 입자가 포획되어 반응 조건과 기술이 잘 작동한다는 것을 보여주는 하나(대조군), 두번째는 특정 포획 분자를 포함하고 분석 대상 분자가 고정된 입자만 포획한다. 이러한 반응 영역을 통과한 후, 유체는 단순히 폐기물 용기의 역할을 하는 최종 다공성 물질인 위크 (wick)로 들어간다. 레터럴 플로우 테스트는 경쟁적으로 또는 중첩하여 분석을 수행할 수 있다.
면역조직화학분석( Immunohistochemical analysis): 본 발명의 일부 실시예에 따라 수행되는 면역분석법은 동종 분석 또는 이종 분석일 수 있다. 동종 분석에서, 면역 반응은 일반적으로 특정 항체 (예를 들어, 항-TRAIL, CRP 및/또는 IP-10 항체), 표지된 분석물 및 표적 샘플을 포함한다. 표지로부터 발생하는 신호는 표지된 분석물에 항체 결합시 직접적으로 또는 간접적으로 변형된다. 면역 반응 및 그들의 검출은 모두 동일한 용액 내에서 수행될 수 있다. 사용될 수 있는 면역 화학적 표지에는 자유 라디칼, 방사성 동위 원소, 형광 염료, 효소, 박테리오파지 또는 조효소가 포함된다.
이종 분석법에서, 시약은 일반적으로 샘플, 항체 및 검출 가능한 신호를 생성하는 수단이다. 상술한 바와 같은 샘플이 사용될 수 있다. 항체는 비드 (단백질 A 및 단백질 G 아가로스 비드와 같은), 플레이트 또는 슬라이드와 같은 지지체상에 고정될 수 있고 액체상에서 항원을 함유하는 것으로 의심되는 표본과 접촉될 수 있다.
특정 실시예에 따라, 항체는 다공성 스트립에 고정되어 검출 부위를 형성한다. 다공성 스트립의 측정 또는 검출 영역은 TRAIL용, CRP용 및 IP-10용의 복수의 부위를 포함할 수 있다. 테스트 스트립은 음성 및/또는 양성 대조군에 대한 부위도 포함될 수 있다.
또는 대조군 부위를 테스트 스트립과는 별도의 스트립상에 배치할 수 있다. 선택적으로, 상이한 검출 부위는 상이한 양의 항체, 예컨대 제 1 검출 부위에서 더 많은 양 및 다음 부위에서 적은 양의 항체를 함유할 수 있다. 테스트 샘플 첨가시, 검출 가능한 신호를 나타내는 부위의 수는 샘플에 존재하는 폴리펩타이드의 양을 정량적으로 나타낸다. 검출 부위는 임의의 적합한 검출 가능한 형태로 구성될 수 있고, 전형적으로 테스트 스트립의 너비에 걸쳐 막대 또는 도트의 형태로 구성될 수 있다.
그 다음, 지지체를 액체상으로부터 분리하고, 지지체상 또는 액체상 중 어느 하나를 신호를 생성하는 수단을 사용하여 검출 가능한 신호를 조사한다. 신호는 샘플 내 분석물의 존재와 관련이 있다. 검출 가능한 신호를 생성하는 수단은 방사성 표지, 형광 표지 또는 효소 표지의 사용을 포함한다. 예를 들어, 검출하고자 하는 항원이 제 2 결합 부위를 함유하는 경우, 그 부위에 결합하는 항체를 검출 가능한 군에 컨쥬게이트시키고, 분리 단계 전에 액체상 반응 용액에 첨가할 수 있다. 고체 지지체상에 검출 가능한 군의 존재는 테스트 샘플에서의 항원의 존재를 나타낸다. 적합한 면역 분석법의 예는 올리고뉴클레오타이드, 면역 블로팅, 면역 형광법, 면역 침강법, 화학 발광법, 전기 화학 발광법 (ECL) 또는 효소 결합 면역 분석법이다.
통상의 기술자는 본 명세서에 개시된 방법을 수행하는데 유용한 다수의 특정 면역 분석법 및 그의 변형물을 잘 알고 있을 것이다. 일반적으로 E. Maggio, Enzyme-Immunoassay, (1980) (CRC Press, Inc., Boca Raton, Fla.)를 참고하였고; U.S. Pat. No. 4,727,022 to Skold et al. titled "Methods for Modulating Ligand-Receptor Interactions and their Application,"U.S. Pat. No. 4,659,678 to Forrest et al. titled "Immunoassay of Antigens," U.S. Pat. No. 4,376,110 to David et al., titled "Immunometric Assays Using Monoclonal Antibodies," U.S. Pat. No. 4,275,149 to Litman et al., titled "Macromolecular Environment Control in Specific Receptor Assays,"U.S. Pat. No. 4,233,402 to Maggio et al., titled "Reagents and Method Employing Channeling," and U.S. Pat. No. 4,230,767 to Boguslaski et al., titled "Heterogenous Specific Binding Assay Employing a Coenzyme as Label." 또한 참고하였다.
항체는 수동 결합과 같은 공지된 기술에 따라, 진단 분석 (예를 들어, 단백질 A 또는 단백질 G 아가로스와 같은 비드, 마이크로스피어, 플레이트, 슬라이드 또는 라텍스 또는 폴리스티렌과 같은 재료로 형성된 웰과 같은 비드)에 적합한 고체 지지체에 컨쥬게이트될 수 있다. 본 명세서에 기술된 항체는 마찬가지로 검출 가능한 표지 또는 방사성 표지 (예 : 35S, 125I, 131I), 효소 표지 (예 : 서양 고추냉이 퍼옥시다아제(horseradish peroxidase), 알칼라인 포스파타제(alkaline phosphatase)) 및 형광 표지 (예 : 플루오레세인(fluorescein), Alexa, 녹색 형광 단백질, 로다민)와 같은 그룹과 컨쥬게이트될 수 있다.
TRAIL 측정용 단클론 항체들은 다음을 포함하고, 이제 제한되지 않는다: 마우스, 단클론 (55B709-3) IgG; 마우스, 단클론 (2E5) IgG1; 마우스, 단클론 (2E05) IgG1; 마우스, 단클론 (M912292) IgG1 kappa; 마우스, 단클론 (IIIF6) IgG2b; 마우스, 단클론 (2E1-1B9) IgG1; 마우스, 단클론 (RIK-2) IgG1, kappa; 마우스, 단클론 M181 IgG1; 마우스, 단클론VI10E IgG2b; 마우스, 단클론MAB375 IgG1; 마우스, 단클론 MAB687 IgG1; 마우스, 단클론 HS501 IgG1; 마우스, 단클론 clone 75411.11 마우스 IgG1; 마우스, 단클론 T8175-50 IgG; 마우스, 단클론 2B2.108 IgG1; 마우스, 단클론 B-T24 IgG1; 마우스, 단클론 55B709.3 IgG1; 마우스, 단클론 D3 IgG1; 염소, 단클론 C19 IgG; 토끼, 단클론 H257 IgG; 마우스, 단클론 500-M49 IgG; 마우스, 단클론 05-607 IgG; 마우스, 단클론 B-T24 IgG1; 랫트, 단클론 (N2B2), IgG2a, kappa; 마우스, 단클론 (1A7-2B7), IgG1; 마우스, 단클론 (55B709.3), IgG 및 마우스, 단클론 B-S23* IgG1, 인간 TRAIL/TNFSF10 MAb (Clone 75411), 마우스 IgG1, 인간 TRAIL/TNFSF10 MAb (Clone 124723), 마우스 IgG1, 인간 TRAIL/TNFSF10 MAb (Clone 75402), 마우스 IgG1.
TRAIL 측정용 항체에는 TRAIL 측정용 단클론 항체 및 다클론 항체가 포함된다. TRAIL을 측정하기 위한 항체는 하기의 단백질을 포함하는 목록으로부터 에피토프를 표적으로 하기 위해 개발된 항체를 포함한다: 마우스 골수종 세포주 NS0 유래된 재조합 인간 TRAIL (Thr95Gly281 수탁번호 P50591), 마우스 골수종 세포주 NS0 유래된 재조합 인간 TRAIL (Thr95Gly281, N-terminal Met 및 6His tag를 가진 수탁번호 P50591), 대장균 유래의 (Val114-Gly281, N-말단 Met 유무 수탁번호 Q6IBA9), 인간 혈장 유래 TRAIL, 인간 혈청 유래 TRAIL, 제 1 아미노산이 위치 85-151 사이에 있고 마지막 아미노산은 위치 249-281에 있는 재조합 인간 TRAIL.
CRP 측정용 단클론 항체의 예는 하기를 포함하고, 이제 제한되지 않는다 : 마우스, 단클론 (108-2A2); 마우스, 단클론 (108-7G41D2); 마우스, 단클론 (12D-2C-36), IgG1; 마우스, 단클론 (1G1), IgG1; 마우스, 단클론 (5A9), IgG2a kappa; 마우스, 단클론 (63F4), IgG1; 마우스, 단클론 (67A1), IgG1; 마우스, 단클론 (8B-5E), IgG1; 마우스, 단클론 (B893M), IgG2b, lambda; 마우스, 단클론 (C1), IgG2b; 마우스, 단클론 (C11F2), IgG; 마우스, 단클론 (C2), IgG1; 마우스, 단클론 (C3), IgG1; 마우스, 단클론 (C4), IgG1; 마우스, 단클론 (C5), IgG2a; 마우스, 단클론 (C6), IgG2a; 마우스, 단클론 (C7), IgG1; 마우스, 단클론 (CRP103), IgG2b; 마우스, 단클론 (CRP11), IgG1; 마우스, 단클론 (CRP135), IgG1; 마우스, 단클론 (CRP169), IgG2a; 마우스, 단클론 (CRP30), IgG1; 마우스, 단클론 (CRP36), IgG2a; 토끼, 단클론 (EPR283Y), IgG; 마우스, 단클론 (KT39), IgG2b; 마우스, 단클론 (N-a), IgG1; 마우스, 단클론 (N1G1), IgG1; 단클론 (P5A9AT); 마우스, 단클론 (S5G1), IgG1; 마우스, 단클론 (SB78c), IgG1; 마우스, 단클론 (SB78d), IgG1 및 토끼, 단클론 (Y284), IgG, 인간 C-Reactive Protein/CRP Biot MAb (Cl 232024), 마우스 IgG2B, 인간 C-Reactive Protein/CRP MAb (Clone 232007), 마우스 IgG2B, 인간/마우스/돼지 C-Reactive Protein/CRP MAb (Cl 232026), 마우스 IgG2A.
CRP 측정용 항체는 CRP 측정용 단클론 항체 및 CRP 측정용 다클론 항체를 포함한다.
CRP 측정용 항체는 하기의 단백질을 포함하는 목록으로부터 에피토프를 표적화하기 위해 개발된 항체를 포함한다: 인간 혈장 유도 CRP, 인간 혈청 유도 CRP, 마우스 골수종 세포주 NS0 유래 재조합 인간 C- 반응성 단백질/CRP (Phe17Pro224 수탁번호 P02741).
IP-10 측정용 단클론 항체의 예는 하기를 포함하고, 이에 제한되지 않는다 : IP-10/CXCL10 마우스 항-인간 단클론 (4D5) 항체 (LifeSpan BioSciences), IP-10/CXCL10 마우스 항-인간 단클론 (A00163.01) 항체 (LifeSpan BioSciences), 마우스 항-인간 IP-10 (AbD Serotec), 래빗 항-인간 IP-10 (AbD Serotec), IP-10 인간 mAb 6D4 (Hycult Biotech), 마우스 항-인간 IP-10 단클론 항체 클론 B-C50 (Diaclone), 마우스 항-인간 IP-10 단클론 항체 클론 B-C55 (Diaclone), 인간 CXCL10/IP-10 MAb Clone 33036 (R&D Systems), CXCL10/INP10 항체 1E9 (Novus Biologicals), CXCL10/INP10 항체 2C1 (Novus Biologicals), CXCL10/INP10 항체 6D4 (Novus Biologicals), CXCL10 단클론 항체 M01A 클론 2C1 (Abnova Corporation), CXCL10 단클론 항체 (M05), 클론 1E9 (Abnova Corporation), CXCL10 단클론 항체, 클론 1 (Abnova Corporation), IP10 항체 6D4 (Abcam), IP10 antibody EPR7849 (Abcam), IP10 antibody EPR7850 (Abcam).
IP-10 측정용 항체는 IP-10을 측정하기 위한 단클론 항체 및 IP-10을 측정하기 위한 다클론 항체를 포함한다.
IP-10을 측정하기 위한 항체는 하기의 단백질을 포함하는 목록으로부터 에피토프를 표적화하기 위해 개발된 항체를 포함한다: 재조합 인간 CXCL10/IP-10, 77 개 아미노산 (aa 22-98) 및 N-말단 His 태그를 포함하는 비-당화 폴리펩타이드 사슬, 인터페론 감마 유도성 단백질 10 (125 aa 길이), 77 아미노산 단편 (22-98)을 함유하고 아미노 말단 헥사히스티딘 태그를 가진 총 8.5 kDa의 분자량을 가진 대장균에서 생산된 IP-10 His Tag 인간 재조합 IP-10, N-말단 Met를 가진 대장균 유래의 인간 IP-10 (Val22-Pro98), 인간 혈장 유래 IP-10, 인간 혈청 유래 IP-10, 제 1 아미노산이 위치 1-22에 있고 마지막 아미노산이 71-98 위치에 있는 재조합 인간 IP-10.
본 명세서에 기재된 폴리펩타이드의 발현값은 절대 발현값, 표준화 발현 및/또는 상대 발현값일 수 있다.
일반적인 과학적 맥락에서, 표준화는 측정 데이터를 다른 표준화된 데이터와 직접 비교할 수 있는 데이터로 변환하는 과정이다. 본 발명과 관련하여, 발현값의 측정은, 예를 들어, 측정된 샘플의 불균등한 분해, 분석마다 상이한 로딩 양 및 기타 다양한 오류에 의해 야기되는 오류가 발생하기 쉽다. 보다 구체적으로, 분석된 샘플은 사람의 실수 및 장비 고장으로 인해 의도한 것보다 더 많거나 적은 생물학적 물질을 포함할 수 있다. 따라서, 동일한 오차 또는 편차가 본 발명의 폴리펩타이드 및 발현이 본질적으로 일정한 대조군에 적용된다. 따라서, 대조군의 발현값으로 TRAIL, IP-10 또는 CRP 발현값을 나누어 몫을 산출하는데, 상기 몫은 기술적 결함 또는 부정확성(시험 목적의 샘플을 파괴하는 주요 오류를 제외하고)으로부터 자유롭고 폴리펩타이드의 표준화된 발현값을 나타낸다. 대조군의 발현값은 서로 다른 샘플에서 동일하기 때문에 이러한 표준화에 유효한 공통 기준점을 구성한다.
특정 실시예에 따라, 각각의 폴리펩타이드 발현값은 동일한 대조군을 사용하여 표준화된다.
각각의 프로토콜은 전형적으로 노이즈에 대한 상이한 신호비를 이끌어 결과적으로 상이한 농도가 측정되기 때문에, 절대 발현값은 사용된 정확한 프로토콜에 의존한다. 보다 구체적으로, 바이오마커의 전반적인 경향은 프로토콜 (예: TRAIL은 바이러스성 감염인 경우 증가하고 박테리아성 감염의 경우 감소함)과 상관없이 보존되지만, 측정 규모는 프로토콜에 좌우된다.
서로 다른 프로토콜을 통해 측정된 단백질의 농도의 이러한 변화는 하기의 실시예 5에서 설명되는 바와 같이, 두 프로토콜의 측정을 상호 관련시키고 변환 함수를 계산함으로써 보상될 수 있다.
전형적으로, 분석되는 샘플은 전혈, 혈청, 혈장, 백혈구 또는 혈액 세포를 포함하는 혈액 샘플이다. 바람직하게는, 샘플은 전혈, 혈청 또는 혈장이다.
참고로, TRAIL및 IP-10 및 CRP는 CSF, 타액 및 상피 세포, 골수 흡인, 소변, 대변, 폐포 세척, 가래를 포함하는 다른 조직 및 샘플에서 높게 발현된다. 따라서, 본 발명의 일부 실시예는 이러한 조직 및 샘플에서 TRAIL, CRP 및 IP-10을 측정하는데 사용될 수 있다.
바람직하게는, 폴리펩타이드의 값은 샘플을 얻은 후 약 24 시간 이내에 측정된다. 또는 폴리펩타이드의 농도는 샘플을 얻은 후 24 시간 이내에 저장이 시작될 때 12 ℃ 이하에서 저장된 샘플에서 측정된다.
폴리펩타이드의 값을 결정하기 위해 테스트가 수행되면, 측정 결과를 포함하는 대상체 특이적 데이터 세트가 선택적으로 생성된다.
대상체-특이적 데이터 세트는 비 휘발성 컴퓨터 판독 가능 매체 상에 컴퓨터 판독 가능 포맷으로 저장될 수 있고, 범용 컴퓨터 또는 전용 회로와 같은 하드웨어 프로세서에 의해 선택적으로 및 바람직하게 액세스된다.
언급된 바와 같이, 테스트 대상체 혈액에서의 폴리펩타이드의 값은 폴리펩타이드의 혈중 농도가 아닌 다른 파라미터에 기초하여 대상체가 이미 박테리아성 감염, 바이러스성 감염 또는 비-박테리아성/비- 바이러스성 질병이 있는 것으로 확인된 경우, 복수 대상체의 혈액에서 동일한 폴리펩타이드의 값과 비교된다. 복수의 대상체의 폴리펩타이드의 값은 검증된 진단과 함께, 본 명세서에서 하기에서 기술하는바와 같이 "그룹 데이터 세트" 또는 "예비 진단된 데이터 세트"라고도 지칭되는 제 2 데이터 세트에 저장될 수 있다.
"비 박테리아성/비 바이러스성 질병"이란 박테리아 또는 바이러스에 의해 유발되지 않는 질병을 지칭한다. 여기에는 급성 심근 경색, 신체 상해, 간질 발작, 염증 질환 등의 질병, 곰팡이 질병, 기생충 질환 등이 포함된다.
본 명세서에서 사용된 “바이러스성 감염”이란, 바이러스에 의해 야기된 질병 및 박테리아 성분을 포함하지 않는 질병을 지칭한다.
예를 들어, 특정 대상체의 박테리아성 감염 가능성을 나타내는 하나 이상의 확률 분류 함수를 계산하거나 또는 특정 대상체의 바이러스성 감염 또는 특정 대상체의 비 박테리아성/비 바이러스성 질병 가능성을 계산하기 위한 목적으로 데이터 세트를 분석하는 방법이 하기 실시예 1에 기재된 바와 같이 수행될 수 있다. 예를 들어 진단은 (i) parainfluenza virus 1, 2, 3, 및 4, coronavirus 229E/NL63, adenovirus A/B/C/D/E, bocavirus 1/2/3/4, influenza virus A 및 B, metapneumovirus, coronavirus OC43, rhinovirus A/B/C, respiratory syncytial virus A 및 B, 및 Enterovirus 검출용 Seeplex®RV15, 또는 (ii) Streptococcus pneumoniae, Haemophilus influenzae, Chlamydophila pneumoniae, Legionella pneumophila, Bordetella pertussis, and Mycoplasma pneumoniae 검출용 Seeplex® PB6와 같은 PCR 진단분석법을 사용하여 서포트될 수 있다.
혈액 배양, 소변 배양 및 대변 배양은 Shigella spp., Campylobacter spp. and Salmonella spp.에 대해 실행될 수 있다; cytomegalovirus (CMV), Epstein-Barr virus (EBV), Mycoplasma Pneumonia, 및 Coxiella burnetii (Q-Fever)에 대한 혈청 검사 (IgM 및 / 또는 IgG)가 실행될 수 있다.
방사선 검사 (예 : 호흡기 감염[LRTI]이 의심되는 환자의 흉부 X 선 촬영)는 흉부 감염을 확인하는 데 사용될 수 있다.
대안적으로 또는 부가적으로 적어도 하나의 숙련된 의사가 진단을 확립하는데 사용될 수 있다.
미리 진단된 대상체에서 폴리펩타이드의 발현값을 측정하는 방법은 상기한 기술한 바와 같다.
바람직하게는, 미리 진단된 대상체에서 폴리펩타이드의 발현값을 결정하기 위해 사용된 동일한 방법을 테스트 대상체에서 폴리펩타이드의 값을 결정하는데 사용한다. 따라서, 예를 들어 미리 진단된 대상체에서 폴리펩타이드의 발현값을 결정하기 위해 면역 분석법이 사용되는 경우, 테스트 대상체에서 폴리펩타이드의 값을 결정하기 위해 면역 분석법이 사용되어야 한다.
혈액 샘플의 유형은 데스트 대상체 및 미리 진단된 대상에체서 동일할 필요는 없다. 본 발명자들은 TRAIL에 대한 혈청 및 혈장 값이 매우 유사하다는 것을 보여줄 수 있었다. 따라서, 예를 들어, 미리 진단된 대상체에서 폴리펩타이드의 발현값을 결정하기 위해 혈청 샘플을 사용한다면, 테스트 대상체에서 폴리펩타이드의 값을 결정하기 위해 혈장 샘플을 사용할 수 있다.
그룹 데이터 세트는 바람직하게는 비 휘발성 컴퓨터 판독 가능 매체상의 컴퓨터 판독 가능 포맷으로 저장되며, 범용 컴퓨터 또는 전용 회로와 같은 하드웨어 프로세서에 의해 선택적으로 및 바람직하게 액세스된다. 두 데이터 세트는 동일한 매체에 저장될 수 있으며 선택적으로 동일한 하드웨어 프로세서에서 액세스할 수 있다.
대상체-특이적 데이터 세트에서, 각 항목은 임의로 그리고 바람직하게는 튜플 (D, L)로 기술될 수 있는데, 여기서 D는 데이터 세트 내의 폴리펩타이드를 나타내고, L은 폴리펩타이드 D의 혈중 값을 나타낸다. 따라서, 상기 데이터 세트는 모든 요소가 폴리펩타이드와 각각의 반응에 의해 뻗어있는 2 차원 공간에서 벡터로 기술될 수 있는 이차원 데이터 세트일 수 있다. 그룹 데이터 세트에서 각 항목은 튜플 (S, G, D, L)로 기술될 수 있고, 여기서 S는 특정 대상체을 나타내며 G는 그룹 데이터 세트에서 대상체 S의 진단을 나타내며 D는 폴리펩타이드를 나타내며 L은 폴리펩타이드의 혈중 값을 나타낸다. 따라서, 예시된 그림은 모든 요소가 대상체, 진단, 폴리펩타이드 및 각각의 반응에 의해 뻗어있는 4 차원 공간에서 벡터에 의해 기술될 수 있는 4 차원 데이터 세트에 관한 것이다. 본 발명의 일부 실시예는 더 높은 차원의 데이터 세트의 사용을 고려한다. 이러한 데이터 세트는 이하 기술한다.
그룹 데이터 세트는 임의로 및 바람직하게는 대상체별 데이터 세트의 하나 이상의 항목, 보다 바람직하게는 모든 항목을 포함할 수 있다. 그룹 데이터 세트가 대상체별 데이터 세트의 모든 항목를 포함하는 실시예에서, 전체 데이터 세트가 하나의 포괄적인 데이터 세트에 포함되기 때문에 2 개의 개별 데이터 세트를 사용할 필요는 없다. 그러나, 그러한 포괄적인 데이터 세트는 선택적으로 및 바람직하게는 분석중인 대상체와 관련된 포괄적인 데이터 세트의 일부분과 다른 대상체와 관련된 포괄적인 데이터 세트의 일부분을 구별하는 방식으로 주석이 달려있다. 본 명세서의 문맥에서, 분석중인 대상체와 관련된 포괄적인 데이터 세트의 일부분은 개별 데이터 세트로서 제공되지 않는 경우에도 대상체별 데이터 세트로 지칭한다. 마찬가지로, 다른 대상체와만 관련된 포괄적인 데이터 세트의 일부분은 별도의 데이터 세트로 제공되지 않는 경우에도 그룹 데이터 세트로 지칭한다.
그룹 데이터 세트는 바람직하게는 많은 대상체의 폴리펩타이드 값을 포함한다 (예를 들어, 적어도 10명의 대상체는 바이러스성 감염이 있는 것으로 미리 진단되고, 적어도 10명의 대상체는 박테리아성 감염이 있는 것으로 미리 진단되고, 적어도 10명의 대상체는 비-박테리아성/비-바이러스성 질병이 있거나 적어도 20명의 대상체가 바이러스성 감염이 있는 것으로 미리 진단 받고, 적어도 20명은 박테리아성 감염이 있는 것으로 미리 진단되고 적어도 20명은 비 박테리아성/비-바이러스성 질병이 있는 것으로 미리 진단된다; 또는 적어도 50명은 바이러스성 감염이 있는 것으로 미리 진단 받고, 적어도 50명은 박테리아성 감염이 있는 것으로 미리 진단받고, 적어도 50명은 비 박테리아성/비 바이러스성 질병이 있는 것으로 사전 진단된다.
그룹별 데이터 세트에는 대상체를 설명하는 추가 데이터가 포함될 수 있다. 추가 데이터를 포함하는 데이터 세트는 분석중인 대상체와 다른 대상체 간의 유사성에 대한 추가 정보를 제공하여 예측 가능성의 정확성을 높이기 때문에 유리할 수 있다.
폴리펩타이드의 값 이외의 다른 유형의 데이터의 대표적인 예는 상기 기술한 바와 같이 종래의 실험실 위험 인자 및/또는 임상 파라미터를 포함 하나 이에 한정되지는 않는다
본 실시예는 폴리펩타이드 및 각각의 값을 제외하고 추가 데이터를 포함하는 대상체별 및 그룹 데이터 세트를 고려한다. 일부 실시예에서, 데이터 세트 중 적어도 하나는 하나 이상의 (예를 들어, 복수의) 다차원 항목을 포함하며, 각각의 항목은 적어도 3 개의 차원을 가지며, 일부 실시예에서는 데이터 세트 중 적어도 하나는 하나 이상의 (예를 들어, 복수의) 다차원 항목을 포함하고, 각각의 항목은 적어도 4 개의 차원을 가지며, 일부 실시예에서는 적어도 하나의 데이터 집합은 하나 이상의 (예를 들어, 복수의) 다차원 항목을 포함하며, 각 항목은 적어도 5 개의 차원을 가지며, 데이터 세트는 하나 이상의 (예를 들어, 복수의) 다차원 항목을 포함하며, 각 항목은 5 개 이상의 차원을 갖는다.
추가적 데이터 세트의 차원은 분석 대상체, 다른 대상체 및/또는 TRAIL, CRP 및 IP-10 이외의 폴리펩타이드 값에 관한 추가 정보를 제공한다.
본 발명의 일부 실시예에서, 추가 정보는 전통적인 실험실 위험 인자, 임상 파라미터, 혈중 화학 물질 및/또는 유전적 프로파일 중 적어도 하나에 속한다.
"전통적인 실험실 위험 인자"는 대상체 샘플에서 분리되거나 유래된 바이오 마커를 포함하며, 현재 임상 실험실에서 평가되고 절대 호중구수 (약칭 ANC), 절대 림프구 수 (약칭 ALC) 백혈구 수 (약칭 WBC), 호중구 % (호중구에 대한 백혈구의 분율 및 Neu (%)로 약칭됨), 림프구 % (림프구에 대한 백혈구의 분율 및 Lym (%)으로 약칭됨), 단핵구 % (단핵구인 백혈구의 분율 및 Mon (%)로 약칭됨), 나트륨 (약칭 Na), 칼륨 (약칭 K), 빌리루빈 (약칭 Bili)과 같은 전통적인 국제 위험 평가 알고리즘에서 사용되는 바이오마커를 포함한다.
바람직하게는, 분석 대상체의 전통적인 실험실 위험 인자 중 적어도 하나는 대상체별 데이터 세트에 포함되며, 하나 이상의 (더욱 바람직하게는 모두) 다른 대상체의 전통적인 연구실 위험 인자 중 적어도 하나는 그룹 데이터 세트에 포함된다. 대상체별 데이터 세트가 전통적인 실험실 위험 인자 중 적어도 하나를 포함할 때 위험 요소는 별도의 항목으로 포함될 수 있습니다. 그룹 데이터 세트가 위험 인자를 포함 할 때, 위험 인자는 선택적으로 및 바람직하게는 대상체별로 포함된다. 따라서, 예를 들어, 그룹 데이터 세트 항목은 튜플 (S, G, D, L {R})로 설명할 수 있고, 여기서 S, G, D 및 L는 상술하였으며 {R}은 대상체 S의 하나이상의 위험 인자이다.
“임상 파라미터"는 연령 (나이), 인종 (RACE), 성별 (성), 핵심 체온 (약칭 "체온"), 증상의 초기 출현 이후의 최대 핵심 체온 ("최대 온도"로 약칭), 증상의 초기 출현 ("증상으로 인한 시간")으로부터의 시간, 임신 또는 가족력 (약칭 FamHX)과 같은 대상체 건강 상태 또는 기타 특성의 모든 비-샘플 또는 비-분석 바이오 마커를 포함한다.
바람직하게는, 분석 대상체의 임상 파라미터 중 적어도 하나가 대상체별 데이터 세트에 포함되고, 하나 이상의 (보다 바람직하게는 모두) 다른 대상체의 임상 파라미터 중 적어도 하나가 그룹 데이터 세트에 포함된다. 대상체별 데이터 세트가 임상 파라미터 중 적어도 하나를 포함할 때, 상기 임상 파라미터는 별도의 항목으로서 포함될 수 있다. 그룹 데이터 세트가 임상 파라미터를 포함할 때, 상기 임상 파라미터는 선택적으로 및 바람직하게는 대상체별로 포함된다. 따라서, 예를 들어, 그룹 데이터 세트 항목은 튜플 (S, G, D, L {C})로 기술될 수 있고, 여기서 S, G, D 및 L는 상술하였고 {C}는 대상체의 임상 파라미터이다.
본 명세서에 사용된 "혈액 화학"은 혈액에 용해되거나 혈액을 포함하는 임의의 모든 물질의 농도 또는 농도들을 의미한다. 이러한 물질의 대표적인 예로는 알부민, 아밀라아제, 알칼라인 포스파타제, 중탄산염, 총 빌리루빈, BUN, C-반응성 단백질, 칼슘, 염화물, LDL, HDL, 총 콜레스테롤, 크레아티닌, CPK, a-GT, 포도당, LDH, 무기 인, 리파아제, 칼륨, 총 단백질, AST, ALT, 나트륨, 트리글리세리드, 요산 및 VLDL를 포함하고, 이에 한정되지 않는다.
일 실시예에 따르면, 분석 대상체의 혈액 화학은 대상체별 데이터 세트에 포함되고, 하나 이상의 (보다 바람직하게는 모두) 다른 대상체의 혈액 화학이 그룹 데이터 세트에 포함된다. 대상체별 데이터 세트가 혈액 화학을 포함할 때, 상기 혈액 화학은 별도의 항목으로서 포함될 수 있다. 그룹 데이터 세트가 혈액 화학을 포함할 때, 상기 혈액 화학은 선택적으로 및 바람직하게는 대상체별로 포함된다. 따라서, 예를 들어, 그룹 데이터 세트 항목은 튜플 (S, G, D, L {C})로 기술될 수 있고, 여기서 S, G, D 및 L는 상술하였고 {C}는 대상체의 혈액 화학이다.
본 발명의 일부 실시예에서, 추가 정보는 개인의 유전적 프로파일에 속한다.
본 명세서에 사용된 "유전적 프로파일"은 다수의 상이한 유전자의 분석을 지칭한다. 유전적 프로파일은 개개인의 전체 게놈에 있는 유전자를 포괄할 수 있으며, 특정 유전자 부분 집합(subset)을 포함할 수 있다. 유전적 프로파일은 게놈 프로파일, 프로테오믹 프로파일, 후성 유전 프로파일 및/또는 전사체 프로파일을 포함할 수 있다.
바람직하게는, 분석 대상체의 유전적 프로파일은 대상체별 데이터 세트에 포함되고, 하나 이상의 (보다 바람직하게는 모두) 다른 대상체의 유전적 프로파일은 그룹 데이터 세트에 포함된다. 대상체별 데이터 세트가 유전적 프로파일을 포함할 때, 상기 유전적 프로파일은 별도의 항목으로서 포함될 수 있다. 그룹 데이터 세트가 유전적 프로파일을 포함할 때, 상기 유전적 프로파일은 선택적으로 및 바람직하게는 대상체별로 포함된다. 따라서, 예를 들어, 그룹 데이터 세트 항목은 튜플 (S, G, D, L {P})로 기술될 수 있고, 여기서 S, G, D 및 L는 상술하였고 {P}는 대상체의 유전적 프로파일이다.
상기 방법은 임의로 및 바람직하게는 폴리펩타이드의 값을 비-휘발성 컴퓨터 판독 가능 매체상에 적어도 일시적으로 저장하는 단계로 진행하며, 상기 판독 가능한 매체로부터 상기 폴리펩타이드의 값을 추출하거나 원하는대로 표시할 수 있다.
두 데이터 세트에 액세스하자마자, 상기 방법은 테스트 대상체를 진단하기 위해 분석 단계로 계속 진행된다.
분석은 특정 대상체가 박테리아성 감염, 바이러스성 감염 또는 비-바이러스성 성, 비-박테리아성 질병을 가질 가능성을 나타내는 하나 이상의 확률 분류 함수 f(δ0,δ1), g(δ0,δ1), h(δ0,δ1)를 계산하는 것이다. 전형적으로, f, g 및 h는 f(δ0,δ1) + g(δ0,δ1) + h(δ0,δ1) = 1의 관계를 만족시킨다. 각 분류 함수는 제 1 좌표 δ0 및 제 2 좌표 δ1의 함수이며, 좌표 δ0 및 δ1 각각은 상술한 바와 같이 발현값의 상이한 조합에 의해 정의된다.
상기 분석은 여러 가지 방법으로 실행될 수 있다.
일 실시예에 따르면, 분석은 하나 이상의 확률 분류 함수를 계산하기 위해 2 중 또는 보다 바람직하게는 3 중 분류기(classifier)를 사용한다.
바람직하게는, 분석은 비-박테리아성 감염 가능성을 부여하기 위해 바이러스성 및 비-바이러스성, 비-박테리아성 질병의 확률을 합산한다. 다른 바람직한 실시예에서, 분석은 감염성 질병의 가능성을 부여하기 위해 바이러스성 및 박테리아성 확률을 합산한다. 그러나 또 다른 바람직한 실시예에서, 분석은 비-바이러스성, 비-박테리아성 질병의 확률을 무시하고, 박테리아성 및 바이러스성 확률의 직접적인 비교를 수행한다. 본 발명의 일부 실시예에 따라 데이터 세트를 분석하기에 적합한 예시적인 해석 함수가 이하 제공된다.
본 발명의 일부 실시예에 따른 데이터 세트의 분석은 머신 러닝(machine learning) 절차를 실행하는 것을 포함한다.
본 명세서에 사용된 용어 "머신 러닝"은 패턴, 규칙성 또는 규칙을 유도하여 사전에 수집된 데이터로부터 적절한 데이터를 생성하거나 의미있는 방식으로 데이터를 기술하도록 구성된 컴퓨터 프로그램으로 구현된 절차를 의미한다.
머신 러닝의 사용은 데이터 세트에 다차원 항목이 포함되어 있을 때 특히 유리하다.
상기 그룹 및 대상체 데이터 세트는 머신 러닝 절차가 데이터 세트를 가장 잘 설명하는 파라미터를 추출할 수 있는 테스트 세트로 사용될 수 있다. 파라미터가 추출되면 감염 유형을 예측하는데 사용할 수 있다.
머신 러닝에서 지도 학습이나 지도되지 않은 학습을 통해 정보를 얻을 수 있다. 본 발명의 일부 실시예에서, 머신 러닝 절차는 지도 학습 (supervised learning) 절차를 포함한다. 지도 학습에서는 학습 시스템의 구조를 최적화하기 위해 전역 또는 지역 목표 함수가 사용된다. 즉, 지도 학습에서는 시스템이 학습을 유도하는데 사용되는 원하는 반응이 있다.
본 발명의 일부 실시예에서, 머신 러닝 절차는 지도되지 않은 학습 절차를 포함한다. 지도되지 않은 학습에서는 일반적으로 목표 함수가 없다. 특히, 학습 시스템에는 일련의 규칙이 제공되지 않는다. 본 발명의 일부 실시예에 따른 지도되지 않은 학습의 한 형태는 데이터 객체가 분명하게 클래스 표지되지 않은 지도되지 않은 클러스터링 (unsupervised clustering)이다.
본 발명의 실시예에 적합한 “머신 러닝” 절차의 대표예는 다음을 포함하고, 이에 한정되는 것은 아니다: 클러스터링, 연관 규칙 알고리즘, 특징 평가 알고리즘, 부분 집합 선택 알고리즘, 서포트 벡터 머신, 분류 규칙, 비용-민감성 분류기, 투표 알고리즘, 스태킹 알고리즘 (stacking algorithms), 베이지안 네트워크, 결정 트리, 신경 네트워크, 인스턴스 기반 알고리즘, 선형 모델링 알고리즘, k-니어리스트 네이버 분석, 앙상블 학습 알고리즘, 확률 모델, 그래픽 모델, 로지스틱 회귀 방법 (다항 로지스틱 회귀 방법 포함), 그라데이션 증가 방법, 특이값 분해 방법 및 주성분 분석. 신경 네트워크 모델 중에서 자기-조직화 맵(self-organizing map)과 적응 공진 이론(adaptive resonance theory)은 일반적으로 지도되지 않은 학습 알고리즘으로 사용된다. 적응 공진 이론 모델은 문제의 크기에 따라 클러스터수가 달라지도록하며 사용자가 경계 파라미터라는 사용자 정의 상수를 사용하여 동일한 클러스터의 구성원 간 유사도를 제어할 수 있도록 한다.
다음은 본 실시예에 적합한 일부 머신 러닝 절차의 개요이다.
연관 규칙 알고리즘은 특징들 중 의미있는 연관 패턴을 추출하는 기술이다.
용어 "연관성(Association)"은, 머신 러닝의 맥락에서, 특정 클래스 또는 숫자 값을 예측하는 기능뿐만 아니라 특징들 간의 상호 관계를 지칭한다. 연관성은 연관 규칙을 찾고, 패턴을 찾고, 특징 평가를 수행하고, 특징 하위 집합 선택을 수행하고, 예측 모델을 개발하고, 특징 간의 상호 관계를 이해하는 것을 포함하지만, 이에 제한되지 않습니다.
"연관 규칙(association rules)"이라는 용어는 데이터 세트 내에서 자주 함께 발생하는 요소를 나타낸다. 여기에는 연관 패턴, 차별 패턴, 빈번한 패턴, 닫힌 패턴 및 거대한 패턴이 포함되나, 이에 제한되지 않는다.
연관 규칙 알고리즘의 일반적인 기본 단계는 모든 관측치 중에서 가장 빈번한 항목 또는 특징 세트를 찾는 것이다. 목록을 얻으면 규칙을 추출할 수 있다.
전술한 자기 조직화 맵은 고차원 데이터의 시각화 및 분석에 자주 사용되는 지도되지 않는 학습 기술이다. 일반적인 응용 프로그램은 맵 상의 데이터 내에서 중앙 종속성을 시각화하는 데 중점을 둔다. 알고리즘에 의해 생성된 맵은 다른 알고리즘에 의해 연관 규칙의 식별 속도를 높이는데 사용될 수 있다. 알고리즘은 일반적으로 "뉴런 (neurons)"이라고 하는 처리 단위의 그리드(grid)를 포함한다. 각 뉴런은 관측이라고 하는 특징 벡터와 연관된다. 맵은 제한된 모델 세트를 사용하여 사용 가능한 모든 관측치를 최적의 정확도로 나타낸다. 동시에 모델은 그리드에서 정렬되어 유사한 모델은 서로 가깝고 서로 다른 모델은 멀리 떨어져 있게 된다. 이 절차를 통해 데이터에서 특징 간 종속성 또는 연관성을 식별하고 시각화할 수 있다.
특징 평가 알고리즘은 특징의 순위를 지정하거나 그들의 영향을 기반으로 하는 특징을 선택한다.
머신 러닝의 맥락에서 "특징"이라는 용어는 하나 이상의 로우(raw) 입력 변수, 하나 이상의 처리 변수 또는 로우 변수 및 처리 변수를 포함하는 하나 이상의 다른 변수들의 수학적 조합을 나타낸다. 특징은 연속적이거나 분리되어 있을 수 있다.
정보 획득은 특징 평가에 적합한 머신 러닝 방법 중 하나이다. 정보 획득의 정의는 트레이닝 인스턴스 모음에서 불순물의 척도인 엔트로피의 정의를 요구한다. 특정 특징의 값을 알면 발생하는 표적 특징의 엔트로피 감소를 정보 획득이라고 한다. 정보 획득은 감염 유형을 설명할 때 특징의 효율성을 결정하는 파라미터로 사용될 수 있다. 대칭 불확실성은 본 발명의 일부 실시예에 따라, 특징 선택 알고리즘에 의해 사용될 수 있는 알고리즘이다. 대칭 불확실성은 특징을 [0,1] 범위로 표준화함으로써 더 많은 값을 갖는 특징에 대한 정보 획득의 편향(bias)을 보상한다.
부분 집합 선택 알고리즘(Subset selection algorithms)은 평가 알고리즘과 검색 알고리즘의 조합에 의존한다. 특징 평가 알고리즘과 마찬가지로, 부분 집합 선택 알고리즘은 특징의 부분 집합을 정렬한다. 그러나, 특징 평가 알고리즘과는 달리, 본 실시예에 적합한 부분 집합 선택 알고리즘은 부분 집합에 포함된 특징들 간의 중복 정도를 고려하여 감염 유형에 가장 큰 영향을 미치는 특징의 부분 집합을 선택하는 것을 목표로 한다. 특징 부분 집합 선택의 이점에는 데이터 시각화 및 이해 촉진, 측정 및 저장 요구 사항 감소, 교육 및 사용 시간 감소, 분류 기능 향상을 위한 산만한 기능 제거가 포함된다.
부분 집합 선택 알고리즘에 대한 두 가지 기본 접근 방식은 작업할 부분 집합에 특징을 추가 (전방 선택)하는 절차 및 현재 특징의 부분 집합에서 삭제 (후방 제거)하는 절차이다. 머신 러닝에서 전방 선택은 같은 이름의 통계적 절차와 다르게 수행된다. 머신 러닝에서 현재 부분 집합에 추가되는 특징은 교차 유효성 검사를 사용하여 하나의 새 특징으로 보강된 현재 부분 집합의 성능을 평가하여 발견된다. 전방 선택에서 교차 검증을 사용하여 각 새 부분 집합의 예상 성능을 평가하면서 나머지 각 특징을 현재 부분 집합에 차례로 추가하여 부분 집합을 구성한다. 현재 부분 집합에 추가될 때 최고의 성능을 발휘하는 특징은 유지되고 프로세스는 계속된다. 남아있는 특징 중 현재 부분 집합의 예측 능력이 향상되지 않으면 검색이 종료된다. 이 절차는 특징의 로컬 최적 세트를 찾는다.
후방 제거도 비슷한 방식으로 실행된다. 후방 제거를 사용하면 특징 세트를 추가로 축소해도 부분 집합의 예측 능력이 향상되지 않으면 검색이 종료된다. 본 실시예는 전방, 후방 또는 양방향으로 검색하는 검색 알고리즘을 고려한다. 본 실시예에 적합한 검색 알고리즘의 대표적인 예는 포괄적인 검색, 그리디 힐 클라이밍(greedy hill-climbing), 부분 집합의 램덤 퍼터베이션(random perturbations), 래퍼 알고리즘(wrapper algorithms), 확률적 레이스 서치(probabilistic race search), 스키마 서치schemata search), 랭크 레이스 서치(rank race search) 및 베이지안 분류자(Bayesian classifier)를 포함하지만 이에 제한되지 않는다.
의사 결정 트리는 결정을 내리기 위해 입력을 고려하는 것과 관련된 논리적 경로를 형성하는 결정 서포트 알고리즘이다.
"의사 결정 트리"라는 용어는 모델 트리, 분류 트리 및 회귀 트리를 포함하되 이에 제한되지 않는 모든 유형의 트리 기반 러닝 알고리즘을 의미한다.
의사 결정 트리를 사용하여 데이터 세트 또는 그 관계를 계층적으로 분류할 수 있다. 의사 결정 트리는 분지 노드(branch node)와 리프 노드(leaf node)를 포함하는 트리 구조를 가지고 있다. 각 분지 노드는 분할 속성의 값에 대해 수행할 속성 (분할 속성) 및 테스트 (분할 테스트)를 지정하고 분할 테스트의 가능한 모든 결과에 대해 다른 노드로 분지한다. 의사 결정 트리의 루트(root)인 분지 노드를 루트 노드(root node)라고 한다. 각각의 리프 노드는 분류 (예를 들어, 그룹 데이터 세트의 특정 부분이 대상체별 데이터 세트의 특정 부분과 일치하는지 여부) 또는 값을 나타낼 수 있다. 또한, 리프 노드는 표현된 분류 (즉, 분류의 정확성)에 대한 신뢰를 측정하는 신뢰도와 같은 표현된 분류에 관한 추가 정보를 포함할 수 있다. 예를 들어, 신뢰도 점수는 매우 낮은 신뢰도 (예 : 표현된 분류의 표시값이 매우 낮음)를 나타내는 점수 0과 매우 높은 신뢰도(예 : 표현된 분류가 거의 확실 함)를 나타내는 1의 점수인, 0에서 1까지의 연속값일 수 있다.
서포트 벡터 머신은 통계적 러닝 이론을 기반으로 하는 알고리즘이다. 본 발명의 일부 실시예에 따른 SVM (support vector machine)은 분류 목적 및/또는 수치 예측을 위해 사용될 수 있다. 분류를 위한 서포트 벡터 머신은 본 명세서에서 "서포트 벡터 분류기(support vector classifier)"로 지칭되며, 수치 예측을 위한 서포트 벡터 머신은 본 명세서에서 "서포트 벡터 회귀(support vector regression)"로 지칭된다.
SVM은 일반적으로 커널 함수(kernel function)로 특징 지어지며, 생성된 SVM이 분류, 회귀 또는 다른 함수를 제공하는지 여부를 결정한다. SVM은 커널 함수의 적용을 통해 입력 벡터를 고차원 형상 공간으로 맵핑한다. 이 공간에서 결정 초표면 (decision hyper-surface, 분리자(separator)라고도 함)을 분류, 회귀 또는 다른 결정 함수를 제공하도록 구성할 수 있다. 가장 단순한 경우 상기 표면은 하이퍼 평면 (선형 분리자라고도 함)이지만 더 복잡한 분리자도 고려되며 커널 함수를 사용하여 적용할 수 있다. 초표면을 정의하는 데이터 포인트를 서포트 벡터라고 한다.
서포트 벡터 분류기는 가장 가까운 데이터 포인트들로부터 분리기의 거리가 가능한 한 먼 분리기를 선택함으로써, 클래스 외부의 객체들과 연관된 특징 벡터 포인트들로부터 주어진 분류 내의 객체들과 연관된 특징 벡터 포인트들을 분리한다. 서포트 벡터 회귀를 위해 허용 가능한 오차 반경을 가진 고차원 튜브가 구성되어 데이터 세트의 오차를 최소화하는 동시에 관련 곡선 또는 함수의 평탄도를 최대화한다. 즉, 튜브는 곡선 또는 표면에 가장 가까운 데이터 포인트들의 집합으로 정의된 핏 곡선(fit curve) 주위의 주머니이다.
서포트 벡터 머신의 장점은 서포트 벡터가 식별되면 나머지 관측을 계산에서 제거할 수 있으므로 문제의 계산 복잡성을 크게 줄일 수 있다. SVM은 일반적으로 트레이닝 단계와 테스트 단계의 두 단계로 작동한다. 트레이닝 단계에서 결정 규칙을 실행할 때 사용할 서포트 벡터 세트가 생성된다. 테스트 단계에서 의사 결정은 결정 규칙을 사용한다. 서포트 벡터 알고리즘은 SVM을 트레이닝하기 위한 방법이다. 알고리즘 실행에 의해, SVM을 특성화하는 서포트 벡터를 포함하는 파라미터의 트레이닝 세트가 생성된다. 본 실시예에 적합한 서포트 벡터 알고리즘의 대표적인 예는 일련적 최소 최적화(sequential minimal optimization)를 포함하고, 이에 제한되지 않는다.
본 발명에 따라 사용될 수 있는 회귀 기술은 선형 회귀(linear Regression), 다중 회귀(Multiple Regression), 로지스틱 회귀(logistic regression), 프로빗 회귀(probit regression), 서수 로지스틱 회귀(ordinal logistic regression), 서수 프로빗-회귀(ordinal Probit-Regression), 푸아송 회귀(Poisson Regression), 음성 이항 회귀(negative binomial Regression), 다항 로지스틱 회귀 (multinomial logistic Regression, MLR) 및 절단된 회귀(truncated regression)를 포함하지만, 이에 제한되지 않는다.
로지스틱 회귀(logistic regression) 또는 로짓 회귀 (logit regression)는 하나 이상의 예측 변수를 기반으로 하는 범주형 종속 변수(제한된 수의 값을 가질 수 있고, 상기 값의 크기는 중요하지 않지만 크기의 순서는 중요하거나 중요하지 않을 수 있는 종속 변수)의 결과를 예측하는데 사용되는 회귀 분석의 한 유형이다. 로지스틱 회귀 분석은 다항식 변형을 포함한다. 다항 로지스틱 회귀 모델은 두 개 이상의 분리된 결과를 허용하여 회귀를 일반화하는 회귀 모델이다. 즉, 독립 변수 세트 (실수값, 이진값, 절대값 등일 수 있음)가 주어지면서 범주별로 분배된 종속 변수의 다른 가능한 결과의 확률을 예측하는데 사용되는 모델이다.
로지스틱 회귀의 장점은 예측 신뢰도의 해석 가능한 척도 즉 확률을 할당한다는 것이다. 예를 들어, 75% 및 99%의 확률로 박테리아성에 감염될 것으로 예측된 환자는 SVM 해석 기능을 사용할 때 각각 박테리아성으로 할당되지만 후자가 더 높은 확률을 가졌다는 사실은 숨겨진다. 가능성의 신뢰값을 할당하면 임상적 판단에 영향을 줄 수 있는 중요한 임상 정보가 추가된다.
중요한 것은 각 환자에 대한 감염 유형의 가능성을 계산하면 상기 시스템으로 하여금 매우 확실하게 분류할 수 없는 환자를 합리적으로 걸러내게 할 수 있다. 이는 도 5에 설명되어 있다. 따라서 결과가 40% 박테리아성 감염 가능성을 할당할 때, “40%”의 점수를 가진 100명의 환자 중 40명은 박테리아성 감염일 것이다.
또한 가능성 점수에 대한 임계값을 사용하여 테스트 대상체의 비-이진법 분류를 할당할 수 있다. 예로서, 30% 이하의 박테리아성 가능성이 있는 테스트 대상체는 낮은 박테리아성 감염 확률이 할당될 수 있다. 30%에서 70% 사이는 중간 박테리아성 감염 확률 및 70% 이상은 높은 박테리아성 감염 확률이 할당될 수 있다. 다른 임계값이 사용될 수 있다.
Least Absolute Shrinkage and Selection Operator (LASSO) 알고리즘은 선형 회귀에 대한 수축 및/또는 선택 알고리즘이다. LASSO 알고리즘은 L1 표준 정규화 (계수들의 절대값의 합에 대한 경계), L2 표준 정규화 (계수의 제곱의 합에 대한 경계) 및 기타 등등이 될 수있는 정규화로 제곱된 오류의 일반적인 합을 최소화할 수 있다. LASSO 알고리즘은 웨이브릿 계수(wavelet coefficient)의 소프트 임계값(soft-thresholding), 전방 단계 회귀(forward stagewise regression) 및 부스팅 방법(boosting method)과 관련될 수 있다. LASSO 알고리즘은 논문 Tibshirani, R, Regression Shrinkage and Selection via the Lasso, J. Royal. Statist. Soc B., Vol. 58, No. 1, 1996, pages 267-288에 설명되어 있고, 이의 개시 내용은 본 명세서에 참고로 포함된다.
베이지안 네트워크(Bayesian network)는 변수 및 변수 간의 조건부 상호 의존성을 나타내는 모델이다. 베이지안 네트워크에서 변수는 노드로 표시되며 노드는 하나 이상의 링크로 서로 연결될 수 있다. 링크는 두 노드 간의 관계를 나타낸다. 노드는 일반적으로 노드가 연결된 다른 노드의 상태가 주어진 노드의 상태 확률을 결정하는데 사용되는 해당 조건부 확률 테이블을 가지고 있다. 일부 실시예에서, 베이즈 최적 분류기 알고리즘(Bayes optimal classifier algorithm)은 그 분류의 확률을 예측하고, 또한 트레이닝 세트로부터 얻어진 다른 가설들 각각으로부터의 확률을 계산하고 이러한 확률을 향후 감염 유형에 대한 가중치로 사용하기 위한 새로운 레코드에 최대 사후 가설(maximum posteriori hypothesis)을 적용하는데 사용된다. 최상의 베이지안 네트워크에 대한 검색에 적합한 알고리즘은 글로벌 스코어 메트릭 기반 알고리즘(global score metric-based algorithm)을 포함하고, 이에 제한되지 않는다. 네트워크 구축에 대한 대안적인 접근 방법으로 마르코프 블랭킷(Markov blanket)이 사용될 수 있다. 마르코프 블랭킷은 노드의 부모, 자식 및 자식의 부모로 구성된 경계 외부의 어떤 노드에 의한 영향을 받지 않도록 노드를 격리한다.
인스턴스 기반 알고리즘(Instance-based algorithms)은 트레이닝 세트에서 생성된 (한 번) 트리 또는 네트워크에 대한 예측을 기반으로하는 대신 각 인스턴스에 대한 새로운 모델을 생성한다.
머신 러닝의 맥락에서 "인스턴스"라는 용어는 데이터 세트의 예를 지칭한다.
인스턴스 기반 알고리즘은 일반적으로 전체 데이터 세트를 메모리에 저장하고 테스트 중인 것과 유사한 일련의 레코드로부터 모델을 작성한다. 이러한 유사성은, 예를 들어, 유클리드 거리(Euclidian distances)를 사용하는 등의 니어리스트-네이버(nearest-neighbor) 또는 로컬 가중 방법(ocally weighted methods)을 통해 평가될 수 있다. 일련의 레코드가 선택되면 최종 모델이 나이브 베이즈(naive Bayes)와 같은 여러 다른 알고리즘을 사용하여 작성될 수 있다.
본 발명은 또한 임의의 수의 세팅으로 환자 또는 대상체 집단을 스크리닝하는데 사용될 수 있다. 예를 들어, 건강 유지 조직, 공중 보건 기관 또는 학교 보건 프로그램은 상술한 바와 같이 중재가 필요한 사람들을 식별하거나 역학 자료 수집을 위해 대상체 그룹을 스크리닝할 수 있다. 보험 회사 (예 : 건강, 생명 또는 장애)는 적용 범위 또는 가격 결정 과정에서 신청자를 심사하거나 가능한 중재을 위해 기존 고객을 심사할 수 있다. 그러한 인구 집단에서 수집된 데이터는 특히 감염과 같은 상태로의 임상 진행과 관련될 때, 예를 들어 건강 유지 조직, 공중 보건 프로그램 및 보험 회사의 운영에 유용할 것이다. 이러한 데이터 어레이 또는 컬렉션은 머신 판독 가능 매체에 저장될 수 있고 개선된 의료 서비스, 비용 효과적인 의료, 개선된 보험 운영 등을 제공하기 위해 임의의 수의 건강 관련 데이터 관리 시스템에서 사용될 수 있다. 예를 들어, 미국 특허 출원 번호 제 2002/0038227호; 미국 특허 출원 제 2004/0122296 호; 미국 특허 출원 제 2004/0122297호; 및 미국 특허 제 5,018,067호에 기재되어 있다. 이러한 시스템은 내부 데이터 저장매체로부터 직접 데이터에 액세스하거나 여기에 자세히 설명 된대로 하나 이상의 데이터 저장 사이트에서 원격으로 액세스 할 수 있다.
머신-판독 가능 저장 매체는 머신 판독 가능 데이터 또는 상기 데이터를 사용하기 위한 명령으로 프로그램된 머신을 사용할 때 다양한 목적으로 사용될 수 있는 데이터 어레이로 인코딩된 데이터 저장 물질을 포함할 수 있다. 본 발명의 바이오 마커의 유효량의 측정 및/또는 그 바이오 마커로부터의 결과의 위험성 평가는 특히 프로그램 가능한 컴퓨터상에서 실행되는 컴퓨터 프로그램으로 구현될 수 있으며, 특히 프로세서, 데이터 저장 시스템 (휘발성 및 비 휘발성 메모리 및/또는 저장 요소), 적어도 하나의 입력 장치, 및 적어도 하나의 출력 장치를 포함할 수 있다.
프로그램 코드는 입력 데이터에 적용되어 위에서 설명한 기능을 수행하고 출력 정보를 생성할 수 있다. 출력 정보는 당업계에 공지된 방법에 따라 하나 이상의 출력 장치에 적용될 수 있다. 컴퓨터는 예를 들어 퍼스널 컴퓨터, 마이크로 컴퓨터, 또는 종래 디자인의 워크스테이션일 수 있다.
각각의 프로그램은 컴퓨터 시스템과 통신하는 높은 수준의 절차적 또는 객체 중심의 프로그래밍 언어로 구현될 수 있다. 그러나 원하는 경우 프로그램을 어셈블리 언어 또는 기계 언어로 구현할 수 있다. 언어는 컴파일된 언어 또는 해석된 언어일 수 있다. 이러한 각각의 컴퓨터 프로그램은 저장 매체 또는 장치 (예를 들어, ROM 또는 자기 디스켓 또는 본 명세서의 다른 곳에서 정의된 다른 것)에 저장될 수 있으며, 장치가 컴퓨터에 의해 판독되어 여기에 설명된 절차를 수행한다.
본 발명의 건강 관련 데이터 관리 시스템은 또한 컴퓨터 프로그램으로 구성된 컴퓨터 판독 가능 저장 매체로서 구현되는 것으로 고려될 수 있으며, 그렇게 구성된 저장 매체는 컴퓨터를 특정 및 사전 정의된 방식으로 동작하도록 하여 여기에 설명된 다양한 기능들 수행하도록 한다.
기록된 결과물에는 분석 결과, 결과, 진단, 예측 및/또는 치료 권장 사항이 포함될 수 있다. 이러한 정보는 예를 들어 기술자, 의사 및/또는 환자에게 전달될 수 있다. 특정 실시예에서, 컴퓨터는 그러한 정보를 환자 및/또는 주치의와 같은 이해 당사자에게 전달하는데 사용될 것이다. 결과물에 기초하여, 대상체에게 적용된 치료는 변경될 수 있다.
일 실시예에서, 결과물(output)은 그래픽으로 표시된다. 다른 실시예에서, 결과물은 수치적으로 (예를 들어, 확률로서) 표시된다. 다른 실시예에서, 결과물은 컬러 인덱스 (예를 들어, 바 디스플레이에서)를 사용하여 생성되며, 여기서 한 색은 박테리아성 감염 및 또 다른 색은 비 박테리아성 감염을 나타낸다. 색상의 강도는 박테리아성 감염/비 감염 가능성과 관련이 있다. 이러한 그래픽 디스플레이는 도 29A-F에 표시된다.
일부 실시예에서, 결과물은 분석이 완료되고 진단 및/또는 예측이 생성된 후 가능한 한 빨리 대상체에게 전달된다. 결과 및/또는 관련 정보는 환자의 담당 의사에 의해 환자에게 전달될 수 있다. 또는 결과물은 서면 보고서 및 전자 메일 또는 전화와 같은 전자 형식의 통신 등 서면을 포함한 임의의 의사소통 수단을 통해 테스트 대상체에게 직접 전달될 수 있다. 전자 메일 통신의 경우와 같이 컴퓨터를 사용하면 통신이 원활해질 수 있다. 특정 실시예에서, 진단 테스트 결과 및/또는 그 테스트에 기초한 결과 및/또는 치료 요구사항을 포함하는 통신이 생성될 수 있고 전기 통신에 숙련된 장인에게 익숙한 컴퓨터 하드웨어 및 소프트웨어 기술의 조합을 사용하여 자동적으로 전달될 수 있다. 건강 관리-지향 통신 시스템의 한 예는 미국 특허 제 6,283,761 호에 설명되어 있다; 그러나, 본 발명은 이러한 특정 통신 시스템을 이용하는 방법들로 제한되지 않는다. 본 발명의 방법의 특정 실시예에서, 샘플의 분석, 질병의 진단 및 분석 결과 또는 진단의 통신을 포함하는 방법 단계의 전부 또는 일부가 다양한 (예를 들어, 외국) 관할 구역에서 수행될 수 있다.
일부 실시예에서, 본 명세서에 기술된 방법은 선택적으로 및 바람직하게는 필수적이지는 않지만, 혈액 내의 폴리펩타이드의 양을 측정하는 제 1구획(예를 들어 면역 조직 화학적 방법을 사용하여) 및 제 1구획에서 측정된 결과를 컴퓨터로 분석하고 진단과 관련된 결과물을 제공하는 제 2구획을 포함하는 적어도 2 개의 구획을 포함하는 휴대용 장치를 포함하는 시스템 (330)을 사용하여 수행된다..
본 발명의 일부 실시예에 따른 시스템 (330)의 대표적인 예에 대한 블록도가 도 34에 도시되어 있다. 시스템 (330)은 휴대용 장치일 수 있지만 필수적이지는 않은 장치 (331)를 포함할 수 있다. 또는, 데스크탑 탑재형 또는 데스크탑 배치형 장치일 수 있는 장치 (331)일 수 있다. 시스템 (330)은 대상체의 혈액 내 폴리펩타이드의 발현값을 측정하도록 구성된 측정 시스템 (333)을 갖는 제 1 구획 (332)을 포함할 수 있다. 측정 시스템 (333)은 자동화된 ELISA, 자동화된 면역 분석 및 자동화된 기능 분석으로 구성된 그룹으로부터 선택된 하나 이상의 자동 분석을 수행할 수 있다. 시스템 (330)은 또한 본 명세서에 설명된 동작 (예를 들어, 제 1 및/또는 제 2 좌표를 정의하는 컴퓨터 프로그램 명령, 곡선 및/또는 평면을 정의하기 위한 컴퓨터 프로그램 명령, 제 1 및/또는 거리를 계산하기 위한 컴퓨터 프로그램 명령, 계산된 거리와 대상체의 박테리아성 및/또는 바이러스성 감염의 존재 유무 또는 감염 가능성을 연관시키는 컴퓨터 프로그램 명령)을 실행하기 위한 컴퓨터 프로그램 명령들을 저장하는 컴퓨터 판독 가능 매체 (338)를 갖는 하드웨어 프로세서 (336)를 포함하는 제 2구획 (334)를 포함할 수 있다. 하드웨어 프로세서 (336)는 제 1 구획 (332)으로부터 발현값 측정치를 수신하고 측정치에 응답하여 프로그램 명령을 실행하도록 구성된다. 선택적으로 및 바람직하게 하드웨어 프로세서 (336)는 처리된 데이터를 디스플레이 장치 (340)에 출력하도록 또한 구성된다.
본 발명의 일부 선택적 실시예에서, 시스템 (330)은 통신 네트워크와 통신한다. 이들 실시예에서, 시스템 (330) 또는 하드웨어 프로세서 (336)는 통신 네트워크 (352)와 통신하는 네트워크 인터페이스 (350)를 포함한다. 도 34에 도시된 바와 같이, 네트워크 (352)는 하드웨어 프로세서 (336)에 의해 수행된 분석 결과 (예를 들어, 대상체의 박테리아성 및/또는 바이러스성 감염 존재 여부 도는 존재할 가능성)를 전송하는데 사용된다. 예를 들어, 시스템 (330)은 분석 결과를 실험실 정보 시스템 (360) 및/또는 시스템 (330)과 같은 복수의 시스템으로부터 데이터를 수집하는 중앙 서버 (362) 중 적어도 하나에 전송할 수있다.
도 39a는 통신 네트워크 (352)가 발현값 측정치를 수신하기 위해 사용되는 실시예에서의 시스템 (330)의 블록도를 나타내는 개략도이다. 이러한 실시예에서, 시스템 (330)은 상술한 바와 같이, 컴퓨터 판독 가능 매체 (338) 및 프로세서 (336)과 같은 하드웨어 프로세서를 포함할 수 있고, 이에 제한되지 않는다. 하드웨어 프로세서 (336)는 네트워크 인터페이스 (350)를 포함할 수 있다. 인터페이스 (350)를 통해, 하드웨어 프로세서 (336)는 측정 시스템 (333)과 같은 측정 시스템으로부터의 발현값 측정치를 수신하고, 수신된 측정값 각각에 응답하여 컴퓨터 판독 가능 매체 (338) 내의 컴퓨터 프로그램 명령을 실행한다. 그 다음 하드웨어 프로세서 (336)는 처리된 데이터를 디스플레이 장치 (340)에 출력할 수 있다.
도 34 및 도 39a에 도시된 실시예의 조합도 고려된다. 예를 들어, 인터페이스 (350)는 네트워크 (352)로부터 발현값 측정치를 수신하고 분석 결과를 네트워크 (352)로 전송하기 위해 사용될 수 있다.
본 발명의 일부 실시예에서, 시스템 (330)은 도 39b의 블록도에 개략적으로 도시된 바와 같이 사용자와 통신한다. 이러한 실시예에서, 시스템 (330)은 상술한 바와 같이 컴퓨터 판독 가능 매체 (338) 및 프로세서 (336)와 같은 하드웨어 프로세서를 포함할 수 있다. 하드웨어 프로세서 (336)는 사용자 (356)와 통신하는 사용자 인터페이스 (354)를 포함한다. 인터페이스 (350)를 통해, 하드웨어 프로세서 (336)는 사용자 (356)로부터 발현값 측정치를 수신한다. 사용자 (356)는 발현값을 외부 소스로부터 얻거나 면역 분석 및 기능 분석으로 구성된 그룹으로부터 선택된 하나 이상의 분석을 수행하거나, 시스템 (333) (도시되지 않음, 도 39a 및 34 참조)를 실행함으로써 얻을 수 있다. 하드웨어 프로세서 (336)는 수신된 측정치들에 응답하여 컴퓨터 판독 가능 매체 (338) 내의 컴퓨터 프로그램 명령들을 실행한다. 그 다음, 하드웨어 프로세서 (336)는 처리된 데이터를 디스플레이 장치 (340)에 출력할 수 있다.
일단 진단이 내려지면 다양한 조치가 취해질 수 있음을 알 수 있다
따라서, 예를 들어, 박테리아성 감염이 판결되면, 대상체는 항생제로 치료받을 수 있다.
항생제의 예는 다음을 포함하고 이에 제한되지 않는다: Daptomycin; Gemifloxacin; Telavancin; Ceftaroline; Fidaxomicin; Amoxicillin; Ampicillin; Bacampicillin; Carbenicillin; Cloxacillin; Dicloxacillin; Flucloxacillin; Mezlocillin; Nafcillin; Oxacillin; Penicillin G; Penicillin V; Piperacillin; Pivampicillin; Pivmecillinam; Ticarcillin; Aztreonam; Imipenem; Doripenem; Meropenem; Ertapenem; Clindamycin; Lincomycin; Pristinamycin; Quinupristin; Cefacetrile (cephacetrile); Cefadroxil (cefadroxyl(; Cefalexin (cephalexin(; Cefaloglycin (cephaloglycin(; Cefalonium (cephalonium(; Cefaloridine (cephaloradine(; Cefalotin (cephalothin); Cefapirin (cephapirin(; Cefatrizine; Cefazaflur; Cefazedone; Cefazolin (cephazolin(;Cefradine (cephradine(; Cefroxadine; Ceftezole; Cefaclor; Cefamandole; Cefmetazole; Cefonicid; Cefotetan; Cefoxitin; Cefprozil (cefproxil(; Cefuroxime; Cefuzonam; Cefcapene; Cefdaloxime; Cefdinir; Cefditoren; Cefetamet; Cefixime; Cefmenoxime; Cefodizime; Cefotaxime; Cefpimizole; Cefpodoxime; Cefteram; Ceftibuten; Ceftiofur; Ceftiolene; Ceftizoxime; Ceftriaxone; Cefoperazone; Ceftazidime; Cefclidine; Cefepime; Cefluprenam; Cefoselis; Cefozopran; Cefpirome; Cefquinome; Fifth Generation; Ceftobiprole; Ceftaroline; Not Classified; Cefaclomezine; Cefaloram; Cefaparole; Cefcanel; Cefedrolor; Cefempidone; Cefetrizole; Cefivitril; Cefmatilen; Cefmepidium; Cefovecin; Cefoxazole; Cefrotil; Cefsumide; Cefuracetime; Ceftioxide; Azithromycin; Erythromycin; Clarithromycin; Dirithromycin; Roxithromycin; Telithromycin; Amikacin; Gentamicin; Kanamycin; Neomycin; Netilmicin; Paromomycin; Streptomycin; Tobramycin; Flumequine; Nalidixic acid; Oxolinic acid; Piromidic acid; Pipemidic acid; Rosoxacin; Ciprofloxacin; Enoxacin; Lomefloxacin; Nadifloxacin; Norfloxacin; Ofloxacin; Pefloxacin; Rufloxacin; Balofloxacin; Gatifloxacin; Grepafloxacin; Levofloxacin; Moxifloxacin; Pazufloxacin; Sparfloxacin; Temafloxacin; Tosufloxacin; Besifloxacin; Clinafloxacin; Gemifloxacin; Sitafloxacin; Trovafloxacin; Prulifloxacin; Sulfamethizole; Sulfamethoxazole; Sulfisoxazole; Trimethoprim-Sulfamethoxazole; Demeclocycline; Doxycycline; Minocycline; Oxytetracycline; Tetracycline; Tigecycline; Chloramphenicol; Metronidazole; Tinidazole; Nitrofurantoin; Vancomycin; Teicoplanin; Telavancin; Linezolid; Cycloserine2; Rifampin; Rifabutin; Rifapentine; Bacitracin; Polymyxin B; Viomycin; Capreomycin.
바이러스성 감염이 판결되면 대상체는 항 바이러스성제로 치료받을 수 있다. 항 바이러스성제의 예는 다음을 포함하지만 이에 한정되지 않는다 : Abacavir; Aciclovir; Acyclovir; Adefovir; Amantadine; Amprenavir; Ampligen; Arbidol; Atazanavir; Atripla; Balavir; Boceprevirertet; Cidofovir; Combivir; Dolutegravir; Darunavir; Delavirdine; Didanosine; Docosanol; Edoxudine; Efavirenz; Emtricitabine; Enfuvirtide; Entecavir; Ecoliever; Famciclovir; Fomivirsen; Fosamprenavir; Foscarnet; Fosfonet; Fusion inhibitor; Ganciclovir; Ibacitabine; Imunovir; Idoxuridine; Imiquimod; Indinavir; Inosine; Integrase inhibitor; Interferon type III; Interferon type II; Interferon type I; Interferon; Lamivudine; Lopinavir; Loviride; Maraviroc; Moroxydine; Methisazone; Nelfinavir; Nevirapine; Nexavir; Oseltamivir; Peginterferon alfa-2a; Penciclovir; Peramivir; Pleconaril; Podophyllotoxin; Raltegravir; Reverse transcriptase inhibitor; Ribavirin; Rimantadine; Ritonavir; Pyramidine; Saquinavir; Sofosbuvir; StavudineTelaprevir; Tenofovir; Tenofovir disoproxil; Tipranavir; Trifluridine; Trizivir; Tromantadine; Truvada; traporved; Valaciclovir; Valganciclovir; Vicriviroc; Vidarabine; Viramidine; Zalcitabine; Zanamivir; Zidovudine; RNAi antivirals; inhaled rhibovirons; monoclonal antibody respigams; neuriminidase blocking agents.
본 명세서에 설명된 방법을 사용하여 수집한 정보는 추가 환자 관리 옵션에 적용될 수 있다. 예를 들어, 정보는 환자가 병원에 입원해야 하는지 또는 입원해서는 안되는지를 결정하는 데 사용될 수 있다. 또한 입원 기간을 연장할지 여부에도 영향을 줄 수 있다. 또한 추가 검사를 수행해야 하는지 여부를 결정하거나 CT 및/또는 X 레이 및/또는 MRI 및/또는 배양 검사 및/또는 혈청 검사 및/또는 특정 박테리아성에 대한 PCR 분석 및/또는 바이러스성에 대한 PCR 분석 및/또는 요추 천자와 같은 절차과 같은 불필요한 검사 수행을 줄일 수도 있다.
환자의 예후, 질병의 중증도 및 결과를 평가하는 것이 종종 임상적으로 유용하다. 본 발명자들은 이제 낮은 레벨의 TRAIL (약 20 pg/ml 또는 약 15 pg/ml 또는 약 10 pg/ml 또는 약 5 pg/ml 또는 약 2 pg/ml)이 환자 나쁜 예후 및 결과 및 높은 질병의 심각성과 유의하게 상관된다는 것을 발견했다. 예를 들어, 본 발명자들은 일반적으로 중증인 중환자실 (ICU)의 성인 환자들은 감염성 또는 비 감염성 여부와 관계없이 병증이 적은 다른 모든 환자와 비교하여 TRAIL 레벨이 현저히 낮다는 것을 발견했다.
따라서, 본 발명의 또 다른 실시예에 따르면, 질병을 갖는 대상체에서 TRAIL 단백질 혈청 레벨을 측정하는 것을 포함하는, 질병에 대한 예후를 예측하는 방법이 제공되며, 여기서 TRAIL 레벨이 미리 결정된 레벨 미만일 때, 예후는 TRAIL 단백질 혈청 레벨이 미리 결정된 레벨 이상인 질병을 갖는 대상체보다 더 좋지 않다.
TRAIL 단백질 혈청 레벨을 측정하는 방법은 상술한 바와 같다.
질병은 감염성 질병 또는 비 감염성 질병일 수 있다. 대상체는 진단되었거나 진단되지 않은 질병이 있을 수 있다.
질병의 특정예는 박테리아성 감염 (예컨대, 균혈증, 수막염, 호흡기 감염, 소변 기관 감염 등), 패혈증, 신체 상해 및 외상, 심혈관 질환, 다발성 기관 부전 관련 질병, 약물-유발 신장독성, 급성 신장 질환, 신장 손상, 진행성 간경화 및 간부전, 급성 또는 만성 좌심실 부전, 우심실 부전 포함/비포함 폐 고혈압 및 다양한 유형의 악성 종양이 포함되고, 이에 제한되지 않는다.
다른 실시예에 따라, 예측의 정확도를 증가시키는데 도움이 되는 추가 폴리펩타이드가 측정된다. 따라서, 예를 들어, 측정될 수 있는 다른 폴리펩타이드는 IP-10, CRP, IL1RA, PCT 및 SAA를 포함한다.
특정 실시 예에 따르면, IP-10, CRP 및 TRAIL이 측정된다.
다른 실시예에 따르면, 단지 TRAIL만이 측정된다.
본 발명자들은 매우 낮은 레벨의 TRAIL (상술한 바와 같이 분류됨)을 갖는 환자는 회복 가능성이 낮고 합병증의 기회가 더 높다는 것을 발견했다. 따라서, 본 발명자들은 대상체가 매우 낮은 레벨의 TRAIL을 갖는 것으로 밝혀지면 최후 수단으로서만 사용되는 제제로 치료되어야 한다고 제안한다.
이러한 제제는, 예를 들어, 완전한 FDA 승인을 받지 않은 실험 제제일 수 있다. 다른 최후의 수단인 제제는 심각한 부작용과 관련이 있는 것으로 알려져 있다. 다른 예시적인 최후의 수단은 반코마이신 (일반적으로 항생제 내성의 확산을 막기 위해 제공되지 않는)과 같은 항생제이다.
일반적으로 최후의 수단으로 간주되지 않는 제제도 또한 제공될 수 있지만, 임상적으로 허용되는 투여량을 초과하는 투여량으로 제공될 수 있다.
본 발명의 이 측면에 따르면, TRAIL 레벨이 미리 결정된 레벨 이상인 경우, 환자는 일반적으로 최후의 수단으로 치료를 받아서는 안된다.
본 발명자들은 건강한 개인 또는 비 감염성 질병 환자의 TRAIL의 기초 레벨은 임신 연령 (t-test P<0.001) 동안 남성과 비교하여 여성에서 더 낮지만 (도 37a 참조), 임신 연령 전 또는 후(t-test P = 0.9, 도 37a)에서 불변임을 발견했다. 이러한 경향은 감염성 질병이 있는 환자에서는 관찰되지 않았다.
통상의 기술자에게 명백한 바와 같이, 이러한 나이 의존적 동역학은 박테리아성, 바이러스성 및 비-감염성 또는 건강한 개인을 구별하는 모델을 개선시키는데 사용될 수 있다.
예를 들어, 상기 모델은 연령 및 성별 파라미터를 포함할 수 있다. 대상체의 연령이 임신이 가능한 특정 범위 (예를 들어, 약 13 내지 45세)이고 대상체가 남성인 경우, 임신 가능 연령의 남성의 TRAIL 모델 계수가 사용될 수 있다. 대상체의 연령이 임신이 가능한 범위 내에 있고 대상체가 여성이면 임신 가능 연령의 여성의 TRAIL 모델 계수를 사용할 수 있다. 대상체의 연령이 임산 가능 범위를 벗어나면 성적 불변성 (gender invariant) 인 TRAIL 모델 계수를 사용할 수 있다.
따라서, 본 발명의 또 다른 측면에 따르면, 임신 가능 연령의 여성 대상체에서 감염 유형을 결정하는 방법이 제공되며, 상기 방법은 대상체의 TRAIL 단백질 혈청 레벨을 임신 가능 연령의 건강한 여성 대상체 또는 임신 가능 연령의 건강한 여성 대상체 집단의 TRAIL 단백질 혈청 레벨에 대응하는 미리 결정된 임계값과 비교하는 단계을 포함하며, 상기 TRAIL 단백질 혈청 수준과 상기 미리 결정된 임계값 간의 차이는 감염 유형을 나타낸다.
따라서, 본 발명의 또 다른 측면에 따르면, 임신 가능 연령의 남성 대상체에서 감염 유형을 결정하는 방법이 제공되며, 상기 방법은 대상체의 TRAIL 단백질 혈청 레벨을 임신 가능 연령의 건강한 남성 대상체 또는 임신 가능 연령의 건강한 남성 대상체 집단의 TRAIL 단백질 혈청 레벨에 대응하는 미리 결정된 임계값과 비교하는 단계을 포함하며, 상기 TRAIL 단백질 혈청 수준과 상기 미리 결정된 임계값 간의 차이는 감염 유형을 나타낸다.
감염 유형을 규정하거나 배제하기 위해 미리 결정된 임계값을 사용할 수 있다.
따라서, 예를 들어 TRAIL 단백질 혈청 레벨이 제 1 미리 결정된 임계값 이상인 경우, 감염 유형은 바이러스성이다.
예를 들어 TRAIL 단백질 혈청 레벨이 제 2 미리 결정된 임계값 이상인 경우, 감염 유형은 박테리아성이다.
예를 들어 TRAIL 단백질 혈청 레벨이 제 3 미리 결정된 임계값 이하인 경우, 감염 유형은 박테리아성이다.
예를 들어 TRAIL 단백질 혈청 레벨이 제 4 미리 결정된 임계값 이하인 경우, 감염 유형은 바이러스성이다.
일반적으로, 본 명세서에 언급된 건강한 남성 또는 여성 대상체는 알려진 질병이 없다. 특정 실시예에 따르면, 대조군 대상체에는 감염성 질병이 없다.
일반적으로, 대상체의 TRAIL 단백질 혈청 레벨과 미리 결정된 임계값 사이의 차이는 상기 설명한 바와 같이 통계적으로 유의한 차이이다.
본 명세서에 사용된 용어 "약"은 10 %를 의미한다.
용어 "포함한다", "포함하는", "함유한다", "함유하는", "갖는" 및 이들의 복합체는 "포함하지만 이에 한정되지 않는다"를 의미한다.
"~으로 구성된"이라는 용어는 "포함하고 제한된"을 의미한다.
용어 "필수적으로 이루어진"의 의미는 추가의 성분, 단계 및/또는 부분이 청구된 조성물, 방법 및/또는 구조의 본질적이고 새로운 특징을 실질적으로 변경하지 않는 한도에서, 상기 조성물, 방법 및/또는 구조가 추가의 성분, 단계 및/또는 부분을 포함할 수 있음을 의미한다
본 발명 전반에 걸쳐, 본 발명의 다양한 실시예는 범위 포맷으로 제공될 수 있다. 범위 포맷에서의 설명은 단지 편의상 및 간략화를 위한 것이며 본 발명의 범위를 제한하여 해석되어서는 안된다. 따라서, 범위의 설명은 가능한 모든 부분의 범위 및 그 범위 내의 개별적인 수치를 구체적으로 개시한 것으로 간주되어야 한다. 예를 들어, 1 내지 6과 같은 범위의 설명은 1 내지 3, 1 내지 4, 1 내지 5, 2 내지 4, 2 내지 6, 3 내지 6 등과 같은 하위 범위를 구체적으로 개시한 것으로 간주되어야 하고 해당 범위 내의 개별 번호 (예 : 1, 2, 3, 4, 5 및 6)를 개시한 것으로 간주되어야 한다. 이는 범위의 폭에 관계없이 적용된다.
본 명세서에 사용된 용어 "방법"은 공지된 방식으로 공지되어 있거나 공지된 방식으로 용이하게 개발된 방식, 수단, 기술 및 절차를 포함하지만 이에 제한되지 않는 주어진 작업을 수행하기 위한 방식, 수단, 기술 및 절차를 의미한다. 화학적, 약리학적, 생물학적, 생화학적 및 의학적 실무자에 의한 기술 및 절차를 의미한다.
본 명세서에서 사용된 용어 "치료하는"은 증상의 진행을 실질적으로 방지, 실질적으로 억제, 지연 또는 반전시키는 것, 증상의 임상적 또는 미적 증상을 실질적으로 완화시키는 것 또는 증상의 임상적 또는 미적 증상의 출현을 실질적으로 방지하는 것을 포함한다.
명확한 설명을 위해, 별도의 실시예들의 문맥에서 설명된 본 발명의 특정 특징들은 또한 단일 실시예에서 조합하여 제공될 수 있다. 반대로, 간략화를 위해, 단일 실시예와 관련하여 기술된 본 발명의 다양한 특징은 또한 개별적으로 또는 임의의 적절한 서브 콤비네이션으로 또는 본 발명의 임의의 다른 설명된 실시예에 적합하게 제공될 수 있다. 다양한 실시예들의 문맥에서 설명된 어떤 특징들은 그 실시예가 이들 요소 없이는 작동하지 않는 한, 그러한 실시예들의 본질적인 특징으로 간주되어서는 안된다.
상술한 바와 같이 청구 범위에 청구된 바와 같은 본 발명의 다양한 실시예 및 측면은 하기 실시예에를 통해 뒷받침 된다.
실시예
상기 실시예와 함께 본 발명의 일부 실시예를 비 제한적으로 설명하는 하기의 실시예를 참조한다.
일반적으로, 본 명세서서 사용된 명명법 및 본 발명에서 이용되는 실험실 과정은 분자, 생화학적, 미생물학적 및 재조합 DNA 기술을 포함한다. 이러한 기술은 문헌에서 상세히 설명된다. 예를 들어, "Molecular Cloning: A laboratory Manual" Sambrook et al., (1989); "Current Protocols in Molecular Biology" Volumes I-III Ausubel, R. M., ed. (1994); Ausubel et al., "Current Protocols in Molecular Biology", John Wiley and Sons, Baltimore, Maryland (1989); Perbal, "A Practical Guide to Molecular Cloning", John Wiley & Sons, New York(1988); Watson et al., "Recombinant DNA", Scientific American Books, New York; Birren et al. (eds) "Genome Analysis: A Laboratory Manual Series", Vols. 1-4, Cold Spring Harbor Laboratory Press, New York (1998); methodologies as set forth in U.S. Pat. Nos.4,666,828; 4,683,202; 4,801,531; 5,192,659 and 5,272,057; "Cell Biology: A Laboratory Handbook", Volumes I-III Cellis, J. E., ed. (1994); "Culture of Animal Cells - A Manual of Basic Technique" by Freshney, Wiley-Liss, N. Y. (1994), Third Edition; "Current Protocols in Immunology" Volumes I-III Coligan J. E., ed. (1994); Stites et al. (eds), "Basic and Clinical Immunology" (8th Edition), Appleton & Lange, Norwalk, CT (1994); Mishell and Shiigi (eds), "Selected Methods in Cellular Immunology", W. H. Freemanand Co., New York (1980)를 참고한다. 이용 가능한 면역 분석법은 특허 및 과학 문헌에서 광범위하게 기술되어 있다, 예를 들어, U.S. Pat. Nos. 3,791,932; 3,839,153; 3,850,752; 3,850,578; 3,853,987; 3,867,517; 3,879,262; 3,901,654; 3,935,074; 3,984,533; 3,996,345; 4,034,074; 4,098,876; 4,879,219; 5,011,771 및 5,281,521; "Oligonucleotide Synthesis" Gait, M. J., ed. (1984); "Nucleic Acid Hybridization" Hames, B. D., and Higgins S. J., eds. (1985); "Transcription and Translation" Hames, B. D., and Higgins S. J., eds. (1984); "Animal Cell Culture" Freshney, R. I., ed. (1986); "Immobilized Cells and Enzymes" IRL Press, (1986); "A Practical Guide to Molecular Cloning" Perbal, B., (1984) and "Methods in Enzymology" Vol. 1-317, Academic Press; "PCR Protocols:A Guide To Methods And Applications", Academic Press, San Diego, CA (1990); Marshak et al., "Strategies for Protein Purification and Characterization - A Laboratory Course Manual" CSHL Press (1996)를 참고한다. 이들 모두는 본 명세서에서 완전히 설명된 것처럼 참고 문헌으로 포함된다. 기타 일반적인 참조는 이 문서 전체에서 제공된다. 그 안의 절차는 당해 기술 분야에서 잘 알려진 것으로 생각되며, 독자의 편의를 위해 제공된다. 여기에 포함된 모든 정보는 참조로 여기에 통합된다.
실시예
1
박테리아 및 바이러스 감염을 구별하기 위한 숙주-
프로데옴
시그니쳐
: 전향적 멀티-센터 관찰 연구
방법
연구 모집단: 총 1002 명의 환자가 이 연구에 참여했다. 소아 환자 (18 세 이하)는 소아 응급실 (PED), 소아과 및 외과로부터, 성인 (18 세 이상)은 응급실, 내과 및 외과에서 모집되었다. 해당되는 경우 각 참가자 또는 법적 보호자로부터 정보 제공 동의를 얻었다. 감염 질병 집단(cohort)에 대한 포함 기준 : 급성 감염 질병의 임상적 의심, 증상 발병 후 > 37.5 ℃의 최고 발열 및 증상 지속 기간이 12 일 이하. 대조군에 대한 포함 기준은 비 감염성 질병 (예 : 외상, 뇌졸중 및 심근 경색) 또는 건강한 대상체의 임상적 인상을 포함한다. 제외 기준 : 등록 전 2 주 동안의 급성 감염 질병 발병의 증거; 진단된 선천성 면역 결핍증; 면역 억제 또는 면역 조절 요법으로 현재 치료; HIV-1, B 형 간염 바이러스 (B 형 간염 바이러스) 또는 C 형 간염 바이러스 (HCV) 감염 (도 1a)이 포함된다. 중요한 것은 광범위한 일반화를 가능하게 하기 위해 등록시 항생제 치료가 연구에서 배제되지 않는다는 것이다.
등록 절차 및 데이터 수집: 각 환자에 대해 인구 통계, 신체 검사, 병력 (주요 불만, 기저 질환, 만성적으로 투여된 약물, 동반 질환, 증상 발현 시간 및 최고 온도), 전 혈구수 (CBC) 화학 패널 (예 : 크레아티닌, 요소, 전해질 및 간 효소)에 대한 정보를 제공한다. 각 환자에게서 추가 미생물 조사를 위해 비강 면봉을 얻었고 단백질 스크리닝 및 검증을 위해 혈액 샘플을 채취했다. 의사가 적절하다고 판단한 추가 샘플을 얻었다 (예 : 요로 감염 [UTI] 의심 환자의 소변 및 대변 샘플, 위장염 [GI]). 방사선 검사는 의사의 재량에 따라 이루어졌다 (예 : 호흡기 감염 [LRTI]이 의심되는 경우 흉부 X 선 검사). 등록 후 30 일 동안 질병 경과 및 치료 반응을 기록했다. 모든 정보는 사용자 전자 사례 보고서 양식 (eCRF)에 기록되었다.
미생물 조사 : 환자들은 비강 면봉 샘플로부터 두가지 multiplex-PCR 진단 검사를 받았다 : (i) parainfluenza virus 1, 2, 3 및 4, coronavirus 229E/NL63, adenovirus A/B/C/D/E, bocavirus 1/2/3/4, influenza virus A 및 B, metapneumovirus, coronavirus OC43, rhinovirus A/B/C, respiratory syncytial virus A 및 B, 및 Enterovirus 검출용 Seeplex®RV15 (n=713); 및 Streptococcus pneumoniae, Haemophilus influenzae, Chlamydophila pneumoniae, Legionella pneumophila, Bordetella pertussis 및 Mycoplasma pneumoniae의 검출용 Seeplex® PB6 (n=633). Multiplex-PCR 분석은 인증된 서비스 연구소에서 수행했다. 환자들은 또한 혈액 배양 (n=420), 소변 배양 (n=188), Shigella spp., Campylobacter spp. 및 Salmonella spp. (n=66) 에 대한 대변 배양 검사; CMV (cytomegalovirus), EBV (Epstein-Barr virus), Mycoplasma Pneumonia 및 Coxiella burnetii (Q-Fever) (n = 167, n = 130, n = 206 및 n = 41)에 대한 혈청 검사 (IgM 및/또는 IgG)를 포함하여 의심되는 임상 증후군에 따라 추가 병원균에 대한 검사를 받았다.
참조 기준 수립 : 명백한 진단, 만장일치 및 대다수 집단 : 엄격한 종합 참조 기준은 진단 정확성 보고 표준 (Standards for Reporting of Diagnostic Accuracy, STARD)의 권고안에 따라 작성되었다. 먼저, 상기 설명한대로 각 환자에 대해 철저한 임상 및 미생물 조사가 수행되었다. 그런 다음, 질병 과정 전반에 걸쳐 수집된 모든 데이터는 3 명의 의사 패널에 의해 검토되었다. 성인 환자 (18 세 이상)의 경우 주치의와 2 명의 감염 질병 전문의가 포함되었고 어린이와 청소년 (18 세 이하)의 경우 소아과 의사, 감염 질병 전문의 및 수석 소아과 의사가 포함되었다. 각 패널 구성원은 각 환자에게 다음 진단 표지 중 하나를 지정했다: (i) 박테리아성; (ii) 바이러스 성; (iii) 명백한 감염 질병이 없거나 건강(대조군); 및 (iv) 불확정. 혼합 감염 (박테리아 + 바이러스) 환자는 유사하게 다뤄지기(예 : 항생제 치료) 때문에 박테리아로 분류되었다. 중요하게는, 패널 위원들은 다른 동료들의 표시와 시그니쳐 결과에 대해 알 수 없다.
이 과정은 진단 확실성의 수준이 증가하는 세 개의 집단을 생성하는 데 사용되었다 (도 1a):
대다수 집단 : 환자들은 세 명의 패널 구성원 중 2명 이상으로부터 동일한 표지를 지정받았다;
만장일치 집단(대다수 집단의 하위그룹): 환자들은 세 명의 패널 구성원 모두로부터 동일한 표지를 지정받았다(상기 용어 “만장일치 집단” 및 “일치된 집단”은 본 명세서에서 교환적으로 사용됨); 및
명확한 진단 집단(만장일치 집단의 하위그룹): 박테리아 감염으로 분류된 환자들은 세 명의 패널 구성원 모두로부터 만장일치로 진단받았고, WBC >15,000/μl (박테리아 감염 위험이 증가한 것으로 나타남) 및 다음 세 가지 미생물학적 확증 중 하나: 균혈증 (양성 혈액 배양), 박테리아성 수막염 신우 신염 (양성 소변 배양 및 신장 관련 초음파 시술), UTI (양성 소변 배양), 패혈성 쇼크 (양성 혈액 배양) 또는 복막 농양 (수술 또는 전산화 단층 촬영으로 입증)을 가졌다. 바이러스성 감염으로 분류된 환자들은 패널 구성원들에 의해 만장일치로 진단되었고 바이러스에 대한 양성 검사 결과가 있었다.
또한 대조군으로 분류된 환자들은 세 명의 패널 구성원 모두로부터 만장일치로 진단되었습니다.
샘플, 절차 및 단백질 측정 : 정맥혈 샘플을 4 ℃에서 5 시간 동안 저장한 후 혈장, 혈청 및 총 백혈구로 분획하고 -80 ℃에서 보관했다. 비강 면봉과 대변 검체는 4 ℃에서 최대 72 시간 동안 보관되었으며 이후 다중 PCR 기반 분석(multiplex PCR-based assay)을 위해 공인된 서비스 연구소로 이송되었다. 스크리닝 단계에서 ELISA (enzyme linked immunosorbent assay), Luminex 기술, 단백질 배열 및 유세포 분석법 (flowly cytometry)을 사용하여 혈청 및 백혈구에서 숙주 단백질을 측정했다. 스크리닝 및 시그니쳐 건축 (Host-proteome screening section 참조) 후, 3 가지 단백질을 선택하여 다음과 같이 측정하였다: CRP는 Cobas 6000, Cobas Integra 400, Cobas Integra 800 또는 Modular Analytics P800 (Roche)을 통해 측정하였다. TRAIL 및 IP-10은 상업적 ELISA 키트 (MeMed Diagnostics)를 사용하여 측정하였다.
통계 분석 : 1 차 분석은 AUC (Receiver Operating Characteristics Curve), 민감도 (TP/P), 특이성 (TN/N), 양성 가능성 비율(LR+ = 민감도/[1- 특이성]), 음성 가능성 비율 (LR- = [1-민감도]/특이성) 및 진단 오즈 비율 (DOR = LR+ / LR-)에 기초한다. 여기서 P, N, TP 및 TN는 양성 (박테리아성 환자), 음선 (바이러스성 환자), 참 양성 (정확하게 진단된 박테리아성 환자), 및 참 음성 (정확하게 진단된 바이러스성 환자)과 각각 대응된다. 통계 분석은 Matlab을 사용하여 수행되었다. 샘플 크기 계산은 하기의 실시예 2에 나타내었다.
숙주-프로테옴 스크리닝 : 다변수 로지스틱 모델의 개발, 트레이닝 및 테스트에 대한 일반적인 개요가 도 1b에 묘사되어 있다. 간략하게, 박테리아성 대 바이러스성 감염 환자의 말초 혈액 샘플에서 차별적으로 발현 될 가능성이 있는 600 개의 단백질 후보자를 확인하는 체계적인 문헌 확인 및 생물 정보학 분석이 수행되었으며, 이 중 일부는 감염에 대한 숙주 면역 반응에서 알려진 역할을 하고 다른 것들은 면역 체계에 직접 연결되지 않았다. 다음으로, 각 단백질 후보는 트레이닝 세트 (50 % 바이러스성 및 50 % 박테리아성)에서 20-30 명의 환자를 측정하고 Wilcoxon rank-sum (WS) P-value <0.01을 사용하여 통계적으로 유의미한 차등 측정으로 단백질을 스크리닝 하였다. 결과적으로 86 개의 단백질이 발견되었다 (FDR(false discovery rate) = 0.07). 이들 단백질 각각을 100 명의 추가 환자 (50 % 바이러스성 및 50 % 박테리아성)에서 평가하고 P<10-4의 t-test 컷오프를 사용하여 추가로 스크리닝하여 바이러스성 대 박테리아성 감염 환자에서 유의하게 차별적으로 발현되는 14 개의 단백질을 얻었다 (FDR <0.001).
시그니쳐 개발 및 검증 : 최적의 단백질 조합을 확인하기 위해 특성 선택 프로세스(feature selection process)를 적용하였다. 특성을 추가하거나 제거하기 위해 일련의 반복을 사용하는 mutual-information min-max 및 forward greedy wrapper의 두 가지 특징 선택 스킴(scheme)이 사용되었다. 이 과정은 트레이닝 세트의 성능 향상이 더 이상 통계적으로 유의하지 않을 때 종료되었다 (P> 0.05). 두 과정 모두 동일한 최종 단백질 3 개에 수렴했다. 단백질 수준을 단일 코어에 통합하기 위해 다중 계산 모델을 조사했다. 이들의 성능은 현저하게 상이하지 않았다 (본 명세서의 하기 실시예 2에서 상세히 설명된 바와 같이 P> 0.1). Multinomial Logistic Regression (MLR) 모델은 환자의 진단에 대한 가능성 점수를 할당하여 확률론적 해석을 제공하기 때문에 선택되었다. 시그니쳐는 이 속성을 사용하여 박테리아성 감염 확률이 중간인 환자를 필터링한다 (0.35 ~ 0.55). '한계 면역 반응 (marginal immune response)'이라는 용어는 박테리아 및 바이러스의 숙주 반응 간의 경계가 존재하기 때문에 이들 환자를 묘사하는데 사용된다. 대다수 집단의 환자들은 트레이닝과 테스트 세트로 나뉘어졌으며, 각각은 환자의 50 %를 구성했다 (도 1b). 트레이닝 세트에는 선별 과정에 참여한 120 명의 환자와 무작위로 배정된 추가 환자가 포함되었다. 테스트 세트에는 나머지 환자가 포함되어 있으며 시그니쳐 성능을 독립적으로 평가하는데 사용되었다. 중요한 것은 테스트 세트 환자 중 누구도 알고리즘을 트레이닝하거나 단백질을 선택하는데 이용되지 않았다. leave-10%-out cross-validation은 모델 성능을 예측하는데 사용되었다. 모델 구성에 대한 더 자세한 내용은 아래의 실시예 2에 나와 있다.
결과
환자 특징: 세 명의 의사가 독립적으로 각 환자(박테리아성, 바이러스성, 대조군 또는 부확실한 중 하나)에게 표지를 할당하였다. 상기 표지들은 향상된 진단 정확성 레벨의 다음의 세 집단(cohorts)을 생성하는데 사용되었다 : 대다수 집단(n=765), 만장일치 집단(n=639) 및 명확한 진단 집단 (n=312)(도 1a). 또한, 의사가 병인을 밝히지 않았거나 대부분의 표지를 받지 못하였기 때문에 98명의 환자들은 불확실한 것으로 표지되었다. 대다수 집단에 대한 상세한 특성은 표 1에 기술되어 있다. 간략하게, 상기 집단은 성별 (여성 47 %, 남성 53 %)과 관련하여 균형이 잡혀있고 소아 환자 56 % (18 세 이하) 및 성인 44 % (18세 초과)를 포함한다. 환자들은 RTI, UTI 및 전신 감염과 같은 광범위한 임상 증후군, 최대 온도 (36-41.5 ℃), 증상 발현 시간 (0-12 일), 합병증 및 약물을 나타낸다 (표 1 및 도 6a-12b). 요컨대, 서양에서 급성 감염병의 대부분을 담당하는 56 종의 병원균이 검출되었다 (도 7a-7b).
표 1 - 대다수 집단 환자들의 기준 특성. 값은 숫자(백분율)이다. 5명 이상의 환자에서 검출된 미생물만을 나타낸다. CNS- 중추 신경계(central nervous system), GI 위장염(gastroenteritis), LRTI 하부 호흡기 감염(lower respiratory tract infection), UTRI 상부 호흡기 감염(upper respiratory tract infection), UTI 요로 감염(urinary tract infection), N/A 데이터가 확보되지 않은 건강한 대조군 또는 환자. 인플루엔자 A 서브그룹은 H1N1 균주를 포함했다. 비정형 박테리아성(atypical bacteria) 서브그룹은 Chlamydophila pneumoniae, Mycoplasma pneumonia 및 Legionella pneumophila을 포함했다. 장 바이러스성(Enteric viruses) 하위그룹은 Rota virus, Astrovirus, Enteric Adenovirus 및 Norovirus G I/II를 포함했다. 임상적 증후군 분석법에서 상기 LRTI 그룹은 폐렴(pneumonia), 기관지염(bronchiolitis), 급성 기관지염(acute bronchitis), 및 후두염(laryngitis)을 포함했고; URTI 그룹은 인두염(pharyngitis), 급성 중이염(acute otitis media), 급성 부비동염(acute sinusitis) 및 급성 편도선염(acute tonsillitis)를 포함했다.
명확한 진단, 만장일치 및 대다수 집단에 대한 시그니쳐 성능 : 600개의 스크리닝된 숙주 단백질 및 그들의 조합 중에서, 대다수 집단 트레이닝 세트에서 박테리아성, 바이러스성 및 대조군 환자를 식별하기 위한 최상의 시그니쳐는 TNF-관련 세포 사멸-유도 리간드 (TNF-related apoptosis-inducing ligand, TRAIL), 인터페론 감마-유도 단백질 10(Interferon gamma-induced protein 10, IP-10) 및 C-반응성 단백질(C-reactive protein, CRP) 세 개의 수용성 단백질을 포함했다(도 2a-2c). 대다수 집단의 테스트 세트에서 박테리아성 및 바이러스성 감염을 식별하기 위한 시그니쳐 AUC는 0.94±0.04 이었다. 전체 대다수 집단에서 10% leave out cross-validation을 사용하여 유사한 결과를 얻었다(AUC=0.94±0.02). 상기 시그니쳐는 스크리닝 단계에서 평가된 모든 개별 단백질들을 능가하였다(P<10-6). 트레이닝 및 테스트 절차가 만장일치 및 명확한 진단 집단에서 반복되었고, 각각 0.96±0.02 및 0.99±0.01의 AUC를 산출하였다. 이러한 성능의 단계적 증가는 세 개의 집단에서의 기준 표준 할당(reference standard assignment)의 확실성의 증가와 일치한다(하기 표 2).
| 모든 환자 | 한계 면역 반응 필터(Marginal immune response filter) | |||||
|
정확성
측정 |
명확한 진단 집단 (Clear diagnosis cohort) |
만장일치 집단 (Unanimous cohort) |
대다수 집단 (Majority cohort) |
명확한 진단 집단 (Clear diagnosis cohort) |
만장일치 집단 (Unanimous cohort) |
대다수 집단 (Majority cohort) |
| AUC | 0.99 (0.98,1.00) |
0.96 (0.94,0.98) |
0.94 (0.92,0.96) |
0.99 (0.98,1.00) |
0.97 (0.95,0.99) |
0.94 (0.92,0.96) |
|
전체
정확성 |
0.94 (0.91,0.97) |
0.90 (0.87,0.92) |
0.88 (0.85,0.90) |
0.96 (0.93,0.99) |
0.93 (0.9,0.96) |
0.91 (0.88,0.94) |
| 민감도 | 0.96 (0.88,1.00) |
0.88 (0.84,0.91) |
0.87 (0.83,0.91) |
0.96 (0.88,1.00) |
0.94 (0.9,0.98) |
0.92 (0.88,0.96) |
| 특이성 | 0.93 (0.89,0.97) |
0.92 (0.89,0.96) |
0.90 (0.86,0.93) |
0.97 (0.89,0.97) |
0.93 (0.9,0.96) |
0.89 (0.86,0.89) |
| LR+ | 13.7 (8,24) |
11.0 (7,16) |
8.7 (6,12) |
32.0 (13,78) |
13.4 (8,21) |
8.4 (6,12) |
| LR- | 0.04 (0.01,0.27) |
0.13 (0.09,0.18) |
0.14 (0.11,0.19) |
0.04 (0.01,0.26) |
0.07 (0.04,0.11) |
0.09 (0.06,0.13) |
| DOR | 319 (43,2383) |
84 (47,150) |
60 (37,98) |
776 (92,6528) |
208 (99,436) |
93 (53,164) |
A. 명확한 진단 (n박테리아성=27, n바이러스성=173), 만장일치 (n박테리아성=256, n바이러스성=271) 및 대다수 (n박테리아성=319, n바이러스성=334) 집단에서 모든 환자들에 대해 leave-10%-out cross-validation를 사용하여 성능을 측정하고 그들의 95% CI를 얻었다. B. 진단의가 시그니쳐를 사용하는 방법과 유사하게 한계 면역 반응(명확한 진단 [n박테리아성=27, n바이러스성=159, n한계=14], 만장일치 [n박테리아성=233, n바이러스성=232, n한계=62] 및 대다수 [n박테리아성=290, n바이러스성=277, n한계=88])으로 환자들을 필터링한 후 상기 분석을 반복하였다.
다음으로, 현재 발명자들은 대다수 집단 테스트 세트에서 감염성(바이러스성 또는 바이러스성) 및 비-감염성 대조군을 식별하기 위한 시그니쳐를 사용하여, 0.96±0.02 AUC를 산출하였다. leave-10%-out cross-validation를 사용하여 추가 평가를 하여 유사한 결과(AUC=0.96±0.01)를 얻었다. 상기 시그니쳐는 개별 단백질 중 어느 것보다 성능이 우수하였다(P<10-8). 또한, 만장일치 및 명확한 진단 집단에서의 평가는 각각 0.97±0.02 및 0.97±0.03으로 향상된 AUC 값을 나타냈다. 시그니쳐 성능에 대한 보존적인 측정을 위해서, 상기 분석은 대다수 집단에 중점을 맞추었다.
실험실 측정, 임상적 파라미터 및 잘-확립된 바이어마커로 비교 : 상기 시그니쳐는 잘 확립된 임상적 파라미터 및 백혈구 수(WBC), 절대 호중구 수(ANC), 호중구 백분율, 최대 온도, 맥박 및 호흡률를 포함하는 실험실 측정으로 비교되었다(도 3a 및 실시예 2). 상기 시그니쳐는 모든 개별 파라미터를 능가하였다(P<10-18). 다음으로 상기 시그니쳐를 일부 임상적 파라미터들의 조합과 비교하였다. 이를 위해 최대 4가지 임상적 파라미터의 모든 조합에 대해 다항 로지스틱 모델을 생성하였다. 가장 좋은 성능을 나타내는 쌍, 트리플렛 및 쿼드로플렛을 도 3a에 도시하였다(다섯번째 파라미터를 추가해도 성능이 향상되지 않음). 상기 시그니쳐는 ANC, 맥박, 림프구 및 % 단핵구 (AUC = 0.94 ± 0.02 vs. 0.77 ± 0.04)로 구성된 가장 좋은 성능을 나타낸 임상 파라미터 조합 (P<10-15)보다 상당히 우수하였다(AUC=0.94±0.02 vs. 0.77±0.04). 다음으로, 패혈증 및 박테리아성 감염을 진단하기 위해 임상 실험에서 일상적으로 사용되는 두 가지 단백질 인 PCT 및 CRP와 상기 시그니쳐 성능을 비교하였다(실시예 2). 상기 시그니쳐는 상기 두 단백질보다 성능이 상당히 우수하였다(각각 P<10-8 및 P<10-6). 또한 상기 시그니쳐는 패혈증 및 박테리아성와 관련된 (예 : TREM, IL-6 및 IL-8), 바이러스성 관련된 (예 : IFN-γ 및 IL-6) 및 염증 관련 (IL-1a 및 TNF-a) 단백질을 포함하는 면역 반응에 확립된 역할을 하는 다양한 범위의 숙주 단백질보다 성능이 더 우수하였다(P<10-8)(도 3b 및 실시예 2).
시그니쳐 성능은 다른 환자 하위그룹간에 강하다 : 실제 임상 환경에 내재되어 있는 환자 및 병원균 이질성은 개별 숙주-바이오마커들의 진단 유용성에 부정적인 영향을 미칠 수 잇다. 상기 시그니쳐, 다중 바이오마커의 조합이 환자 대 환자 변이성에도 불구하고 안정된 성능을 유지할 수 있는지 여부를 조사하기 위해 하위그룹 분석을 수행하였다. 상기 시그니쳐는 성별, 임상적 증후구, 증상 발현 시간, 최대 온도, 병원균 종, 합병증, 만성 질병에 대한 약물 처방 및 임상 부위를 포함하는 넓은 범위의 환자의 특징간에 강하다(0.87 및 1.0 사이의 AUC)(도 4 및 실시예 2). 상기 시그니쳐는 또한 전문가 패널의 만장일치 표지를 받았지만 기술적으로 제외된 환자들의 하위그룹에서 테스트되었고, 0.96±0.06(n박테리아성=27, n바이러스성=14) AUC를 산출하였다. 이것은 상기 시그니쳐가 초기에 제외(예, 발열 환자)된 상태에 보다 광범위하게 적용될 수 있음을 시사한다.
시그니쳐의 성능은 잠재적 콜로나이저(colonizer)의 존재에 의해 영향을 받지 않는다 : 많은 질병을 유발하는 박테리아성도 자연 식물군의 일부이며 증상이 없는 대상체에서 흔히 발견된다. 이러한 박테리아성는 단지 진단만으로 질병의 원인이 될 수는 없기 때문에 상당한 진단적 도전을 제기한다. 따라서 적절한 치료법이 명확하지 않을 수 있다. 본 발명자들은 시그네쳐 성능이 그들의 존재에 의해 영향을 받는지를 질문하였다.
나살 스왑(nasal swabs)에서 PCR에 의해 검출된 Streptococcus pneumoniae (SP) 및 Haemophilus influenzae (HI)은 대다수 집단에서 가장 흔한 두 종의 박테리아성였다(표 1). 박테리아성 및 바이러스성 환자 모두에서 SP 및 HI가 높은 비율로 발견되어(SP: 36% 및 47%; HI: 20% 및 32%), 이들의 존재가 반드시 질병을 유발하지는 않는다는 사실이 입증되었다. 환자들이 SP(SP+: n박테리아성=116, n바이러스성=157; SP-: n박테리아성 =203, n바이러스성 =177)를 가지는지 여부에 따라 계층화되었고 두 그룹의 AUC 성능을 비교하였다. 상당한 차이가 발견되지 않았다(0.93±0.03 vs. 0.94±0.02, P=0.31). HI의 존재 여부는 시그니쳐 성능에 영향이 없었다(0.94±0.04 vs. 0.93±0.02; HI+: n박테리아성=63, n바이러스성=106; HI-: n박테리아성 =256, n바이러스성 =228, P=0.34). 이는 상기 시그니쳐가 SP 및 HI의 존재에 영향을 받지 않음을 나타낸다.
검토
임상 데이터 수집, 화학 패널 및 광범위한 미생물학적 테스트를 포함하는 엄격한 합성 기준 표준 전략이 구성되었고 세 명의 독립적인 의사에 의해 분류되었다. 이 절차는 크기가 감소하고 기준 표준의 확실성이 증가한 세 가지 하위 집단의 분류 체계를 생성하였다: 대다수, 만장일치 및 명확한 진단. 각각의 시그니쳐 AUC는 0.94 ± 0.02, 0.96 ± 0.02 및 0.99 ± 0.01이었다. 이러한 성능에서의 단계적 향상은 기준 표준 확실성 증가에 기여할 수 있다. 그러나 특히 명확한 진단 집단에서의 증가된 정확성은 부분적으로 심각한 질병 또는 직접적인 진단을 받은 환자의 선택 편향 때문일 수 있다. 따라서 명세서에 개시된 주요 분석은 더 넓은 범위의 병의 심각성과 진단하기 어려운 경우를 포착하는 대다수 집단에 초점을 두었다. 이 집단은 잠재적으로 일부 잘못된 표지가 포함되어 있어 시그니쳐 정확도의 보존력이 있는 평가가 가능하다.
상기 시그니쳐는 현재의 미생물학적 테스트에 대한 몇 가지 문제를 다룬다. (i) 접근이 불가능하거나 알려지지 않은 감염 부위를 진단하는 어려움. 상기 시그니쳐는 하부 호흡기 감염 (AUC 0.95 ± 0.03, n = 153) 및 원인없는 발열 (AUC = 0.97 ± 0.03, n = 123)을 포함하는 그러한 사례를 정확하게 진단했다. (ii) 결과를 얻는데 걸리는 기간 연장 (수 시간에서 수 일). 상기 시그너쳐는 가용성 단백질을 측정하여 병원에서 비치된 자동화된 면역 측정 장비와 현장 진단(point-of-care) 장비에서 신속한 측정 (몇 분 이내)이 가능하다. (iii) 혼합된 감염은 바이러스성의 검출이 박테리아성 공동 감염을 배제하지 않기 때문에 진단의 불확실성을 초래할 수 있다. 상기 시그니쳐는 혼합 감염과 순수 박테리아성 감염을 분류하여 이 문제를 해결함으로써 항생제 치료와 관련하여 두 그룹 모두를 유사하게 관리하도록 촉구한다. 혼합된 공동 감염이 반응의 혼합보다는 오히려 순수한 박테리아성와 유사한 프로테옴 숙주 반응을 유도한다는 사실은 바이러스성보다 박테리아성의 경로 우위를 나타낼 수 있다. (iv) 특히 PCR, 미생물학적 테스트의 중요한 단점은 비 박테리아성 질병이 있는 대상체에서 잠재적 콜로나이저의 검출이다. 상기 시그니쳐는 성능은 잠재적 콜로나이저의 존재 여부에 영향을 받지 않았다.
PCT, CRP 및 IL-6와 같은 숙주 단백질은 임상 증상, 혈구 수 및 미생물학에 대한 추가 정보를 전달하기 때문에 박테리아성 감염 진단에 도움을 주기 위해 일상적으로 사용된다. 그러나 환자 및 병원균 사이의 변이성으로 인해 유용성이 제한된다. 숙주 단백질의 조합은 이것을 극복할 수 있는 잠재력을 가지지만 개별 단백질에 비해 미미한 수준의 진단 개선을 가져왔다. 이러한 미미한 개선은 동일한 인자에 민감하여 서로 보상하는 능력이 적은 박테리아성 유래 단백질의 조합이 원인일 수 있다. 따라서, 숙주-단백질, 임상 파라미터 및 다른 테스트를 포함하는 조합에서 더 큰 개선이 관찰되었다. 그러나 이러한 다중 파라미터를 실시간으로 얻는 것은 자주 가능하지 않다.
이를 해결하기 위해 보완적인 특성을 보이는 단백질의 조합이 확인되었다. 구체적으로, TRAIL은 바이러스에 반응하여 유도되고 박테리아에 의해 억제되고, IP-10은 박테리아성 감염보다 바이러스성에서 높고, CRP는 바이러스성 감염보다 박테리아성에서 더 높은 것으로 확인되었다. 박테리아성 감염을 제안하기 위해 향상된 CRP의 유용성은 잘 확립되어 있지만, 일상적으로 사용되는 박테리아성에 의해 유도된 단백질을 보완하기 위한 새로운 바이러스성 유도 단백질의 포함은 증상 발현 시간, 병원균종 및 기타 합병증을 포함하는 넓은 범위의 하위 그룹에 대한 시그니쳐의 견고성에 실질적으로 기여했다. 예를 들어, 소아에서 급성 감염의 5 % -15 %를 일으키는 바이러스성의 중요한 하위 그룹인 아데노바이러스는 박테리아성 감염과 유사한 임상 증상을 유발하기 때문에 진단하기가 특히 어렵다. 일반 실험실 파라미터는 시그니쳐와 비교하여 이러한 하위 그룹에 대에 불충분히 수행된다.(AUC = 0.60±0.10 [WBC], 0.58±0.10 [ANC], 0.88±0.05 [시그니쳐]; n = 223).
감염성 질병 진단의 발전에도 불구하고 박테리아성 감염의 적시의 확인은 여전히 어려움을 겪고 있으며, 심각한 건강 및 경제적 결과로 인해 항생제 오용이 발생한다. 보다 나은 치료 지침에 대한 필요성을 해결하기 위해, 본 발명자는 박테리아성 감염 및 바이러스성 감염을 구별하기 위한 신규 및 전통 숙주-단백질을 조합한 시그니쳐를 개발하고 검증하였다. 큰 표본 크기의 환자에서의 이 발견은 이 숙주-시그니쳐가 임상의가 급성 감염성 질병 환자를 관리하고 항생제 오용을 줄이는 데 도움이 될 가능성이 있음을 시사한다.
실시예
2
박테리아성 및 바이러스성 감염을 구별하기 위한 숙주-프로테옴 시그니쳐 : 전향 적 멀티 센터 관측 연구 - 보충 자료
정확성의 측정: 상기 시그니쳐는 대상체에서 측정한 세가지 단백질 바이오마커의 레벨을 통합하고 박테리아성 대 바이러스성 감염의 가능성을 반영하는 점수를 계산한다. 상기 시그니쳐의 진단 정확성을 정량화하기 위해, 상기 점수의 컷오프를 사용하여 다음 측정에 적용하였다 : 민감도(Sensitivity), 특이성(specificity), 양성 예측도 (positive predictive value, PPV), 음성 예측도 (negative predictive value, NPV), 전체 정확도(total accuracy), 양성 가능성비 (positive likelihood ratio, LR +), 음성 가능성비 (negative likelihood ratio, LR -) 및 진단상의 오즈비 (diagnostic odds ratio, DOR). 이러한 측정은 다음과 같이 정의된다 :
P, N, TP, FP, TN, FN은 각각 양성, 음성, 참-양성, 거짓-음성, 참-음성 및 거짓-음성이다. 유병률(Prevalence)는 양성 클래스의 상대적 빈도이다(즉, 유병률 = P/(P + N)). 달리 언급되지 않는한, 양성 및 음성은 각각 박테리아성 및 바이러스성 감염 환자를 지칭한다.
AUC는 또한 다른 진단방법과 비교하여 독립적인 컷오프를 수행하는데 사용된다. AUC의 방정식 및 신뢰 구간 (CI) 계산에 대한 상세한 내용은 Hanley 및 McNeil를 참조한다. 본 명세서 전체에 걸쳐 정확도 수치가 95% CI로 보고된다.
샘플 크기: 주요 연구 목적은 바이러스성 및 박테리아성 병인을 가진 환자를 분류하기 위한 시그니쳐의 성능을 얻는 것이었다. 전체 집단에 대한 민감도와 특이성가 유의 수준 1 %, P0 = 75 %, 15 %의 차이에 대한 90 %의 힘보다 낮다는 귀무 가설을 기각하는데 필요한 표본 크기 P1 - P0 ≥15 %), 이는 394 명의 환자 (197 바이러스성 및 197 박테리아성)를 산출했다. 또한 약 15 %의 환자가 불확실한 감염원을 갖게 될 것이며, 기술적인 이유 때문에 10 %가 제외될 것이며 10 %는 건강 또는 비 감염성 대조군이 될 것으로 예상된다. 이 연구 결과를 종합해 볼 때 최소한 607 명의 환자 모집이 필요했다. 이 요구 사항은 1002 명의 환자가 모집되었기 때문에 충족되었다.
계산 모델 로지스틸 모델을 구성: 단백질 레벨을 단일 예측 점수로 통합하기 위해 인공 신경망 (ANN), SVM (Support Vector Machines), BN (Bayesian Networks), K-Nearest Neighbor (KNN) 및 MLR (Multinomial Logistic Regression)을 포함하는 다중 계산 모델이 심사되었다. leave-10 % out 교차 검증을 사용하여 대다수 집단에서 얻은 박테리아성 감염 및 바이러스성 감염을 구별하는 AUC는 0.93 ± 0.02 (ANN), 0.93 ± 0.02 (SVM [linear]), 0.94 ± 0.02 [SVM (radial basis function)], 0.92 ± 0.02 (BN), 0.91 ± 0.02 (KNN) 및 0.94 ± 0.02 (MLR)이었다. ANN, SVM 및 MLR 모델 (AUC를 비교할 때 P> 0.1)의 성능에서 유의한 차이는 관찰되지 않았다. 본 발명자들은 MLR을 사용하기로 결정하였는데, 이는 MLR이 환자의 진단에 가능성 점수를 할당함으로써 확률론적 해석을 제공하기 때문이다.
본 발명자들은 박테리아성 및 비 박테리아성 병인을 구별하기 위해 MLR 시그니쳐를 연구하고 테스트하였다. 근본적인 병인의 유병률은 상이한 임상 환경에 따라 다양하기 때문에, 모델 프라이어(priors)를 동일한 기준 유병률(50 % 박테리아성 및 50 % 비-박테리아성)을 반영하도록 조정하였다. 비 박테리아성 그룹 내에서 급성 감염이 의심되는 대상자들 중 바이러스성 대 비-감염성 환자의 예상 유병률을 반영하여 상기 프라이어를 45 % 바이러스성 및 5 % 비-감염성으로 조정하였다. MLR 가중치 및 각각의 95 % 신뢰 구간 및 각 계수와 관련된 p-value는 아래의 표 3-4에 요약하였다. 박테리아성 대 바이러스성 감염 분석에서 확률은 최대 1로 조정되었다(Pb _adjusted=[Pb + Pv] and Pb _adjusted=[Pb + Pv], 여기서 Pb 및 Pv는 각각 박테리아성 및 바이러스성 감염의 확률에 해당한다).
| 제 1 좌표 δ0 (바이러스성) |
제 2 좌표 δ1 (박테리아성) |
|
| Constant | a 0 = -1.299 ± 0.651 | b 0 = -0.378 ± 0.732 |
| TRAIL | a 1 = 0.0088 ± 0.0064 | b 1 = -0.020 ± 0.0084 |
| CRP | a 2 = 0.0605 ± 0.0145 | b 2 = 0.0875 ± 0.015 |
| IP-10 | a 3 = 0.0053 ± 0.0014 | b 3 = 0.0050 ± 0.0014 |
| Class (바이러스성) | Class (박테리아성) | |
| Constant | <0.001 | <0.001 |
| TRAIL | 0.008 | <0.001 |
| CRP | <0.001 | <0.001 |
| IP-10 | <0.001 | <0.001 |
로지스틱 보정 곡선: MLR 모델의 유효성을 평가하기 위해 계산된 예측 확률을 실제로 관찰된 결과와 비교하였다 (도 5). 예측된 확률은 실제 관찰된 확률과 매우 유사하여 모델의 유효성을 더 보여준다.
이 연구에서 사용된 환자 집단 요약 : 총 1002 명의 환자가 모집되었고 892 명이 등록되었다 (110 개가 사전 결정된 제외 기준에 따라 제외되었다). 실시예 1의 '방법' 부분에 기술된 참조 표준 프로세스에 기초하여, 환자들는 4 개의 상이한 진단 그룹에 배정되었다 : (i) 박테리아성; (ii) 바이러스성; (iii) 명백한 감염 질병이 없거나 건강(대조군) 및 (iv) 불확실. 혼합 감염 (박테리아성 + 바이러스성) 환자는 박테리아성 환자와 유사하게 (예 : 항생제 치료를 받음) 관리되기 때문에 박테리아성로 분류되었다 (도 1a). 전체 등록 환자의 89 %가 진단을 받았으며, 문헌에 기록된 한계에 근접한 비율을 보였다. 다음 섹션에서는 최종 진단을 받은 모든 환자를 포함하는 환자 특성에 대해 자세히 설명한다(n = 794 ) : 대다수 집단 환자 765 명과 스크리닝 단계에서 혈청 샘플이 고갈된 29 명의 환자 (도 1a-1b).
연령과 성별 분포: 모든 연령대의 환자가 연구에 모집되었다. 진단된 환자 (진단된 환자, n = 794)에는 성인 (18 세 이상) 환자보다 소아과 (18 세 이하)가 더 많았다(445 명 (56 %) 대 349 명 (44 %)). 연령 분포는 20-80 세의 환자의 경우 비교적 일정했고 소아 환자의 경우 4 세 미만에서 정점에 이르렀다 (도 6a-6b). 소아 환자에서 관찰된 연령 분포는 예상한 것과 일치하며 입원 환자 셋팅(예 : 응급실 [ED], 소아과 및 내부 부서)에서의 배경 분포를 나타낸다. 두 성별 환자가 연구에 모집되었다. 상기 환자 분포는 성별 분포와 관련하여 균형을 이뤘다 (여성 47 %, 남성 53 %).
검출된 병원균 : 병원균 검출율을 최대화하기 위해 다양한 미생물학적 도구가 사용되었다. 급성 감염 질병 환자의 65 %에서 적어도 하나의 병원체가 검출되었다 (진단된 794 명의 환자 중 56 %). 다중 PCR, 항원 검출 및 혈청 검사를 이용하여 총 36 개의 병원균이 검출되었다. 표준 배양 기술 또는 인하우스 PCR을 사용하여 추가 20 병원체가 검출되었다. 모든 주요 병원성 하위 그룹에서 56 개의 다른 병원체가 검출되었다 (도 7a). 이 병원균 확인률은 이전에 발표된 연구에서 보고된 것과 비슷하며 모든 주요 병원성 하위 그룹 (그람 음성균, 그람 양성균, 비정형 박테리아성, RNA 바이러스성 및 DNA 바이러스성)의 병원균을 포함한다. 환자의 13 %에서 앞서 언급한 병원성 하위 집단 중 하나 이상의 병원균이 검출되었다 (도 7a).
이 연구에서 발견된 병원성 균주는 서양의 급성 감염 질병의 대부분을 담당하고 있으며 인플루엔자 A/B, 호흡기 세포 바이러스성 (RSV), 파라인플루엔자, 대장균, A 군 연쇄상구균 등 주요 병원균을 포함한다. 특히, 검출된 병원균의 분석 결과 상기 병원균은 우성이 아님이 드러났다 (도 7b).
관련 생리학적 시스템 및 임상 증후군 : 감염성 질병 환자 (진단된 모든 환자[n=794] (비 감염성 질환자 또는 건강한 대상자 제외, n = 673))는 다양한 생리적 시스템에서 감염을 나타냈다 (도 8). 가장 흔한 생리학적 시스템은 호흡기 (46 %), 그 다음 전신 감염 (22 %)이었다. 상기 언급된 시스템을 포함하지 않고 위장관, 비뇨기, 심혈관 또는 중추 신경계 (CNS) 감염이 아닌 모든 감염은 '기타'(예 : 봉와직염(cellulitis), 농양(abscess))로 분류되었다. 관찰된 생리학적 시스템의 분포는 자연 분포를 나타내며 일년 내내 샘플링된 환자들의 대규모 집단에 대해 보고된 것과 일치한다.
본 연구에서 진단된 환자 (n = 794)는 연중 수집된 소아 및 성인 환자 집단에서 예상되는 임상적 이질성을 반영하는 다양한 임상 증후군 (도 9a-9b)을 나타냈다. 가장 흔한 임상 증후군은 주로 폐렴, 기관지염, 세기관지염, 만성 폐색성 폐 질환 (COPD) 악화 및 비 특이성 LRTI를 포함한 LRTI (21 %)였다. 두 번째로 흔한 증후군은 주로 원인불명 발열 또는 보이지 않는 균혈증을 포함하는 전신 감염 (19 %)이었다. 전신 감염은 주로 3 세 미만의 소아에게서 발견되었지만 일부 성인 환자에서도 발견되었다. 전신 감염은 환자 위험과 테스트/치료 비용 간의 균형이 불분명하기 때문에 진정한 임상적 도전 과제이다. 세 번째로 흔한 임상 증후군은 주로 급성 편도선염, 급성 인두염, 비 특이성 요로 감염, 급성 부비동염 및 급성 중이염을 포함한 URTI (19 %)였다. 다음으로 가장 흔한 증후군은 위장염 (12 %), UTI (7 %), 봉와직염 (4 %)이었다. CNS 감염 (2 %)에는 패혈증 및 무균 성 수막염이 포함되었다. 추가 임상 증후군 (1 %)은 '기타'로 분류되었으며 덜 일반적인 감염 (예: 외이염(otitis externa), 부고환염(epididymitis) 등)이 포함되었다. 임상 증후군 분포에서 관찰된 패턴은 가장 빈번하게 임상적으로 관련된 증후군의 대부분을 나타내며 이전에 발표된 대규모 연구와 일치한다.
중심 체온(Core body temperature): 중심 체온은 감염 질병의 심각성을 평가할 때 중요한 파라미터이다. 최대 체온의 분포는 가장 높게 측정된 체온 (구강(per-os) 또는 직장(per-rectum))을 사용하여 진단된 환자 (n = 794)모두로부터 검사되었다. 최대 체온 분포는 38 ℃에서 40 ℃ 사이에서 상대적으로 균일했으며 39 ℃에서 최고치를 보였다 (도 10). 체온이 <37.5 ℃는 환자의 15 % (비 감염 질병 환자 또는 건강한 환자의 하위 그룹)에서 보고되었다. 체온 ≥40.5 ℃는 드물었다 (환자의 3 % 미만). 전체적으로, 관찰 된 분포는 임상 환경에서 온도의 정상 범위를 나타낸다.
증상 발현 시간 : '증상으로부터의 시간'은 첫 번째 증상의 출현 (첫 번째 증상은 발열일 수 있지만 발열 전의 구토 또는 두통과 같은 다른 증상일 수도 있음)의 지속 시간(일)으로 정의되었다. 각 집단 (진단된 모든 환자, n = 794)에서 증상으로부터의 시간 분포는 조만간 의료 지원을 받을 상당수의 환자가 증상의 시작 후 2-4 일 (환자의 35 %)에서 절정에 달했다 (도 11).
합병증 및 만성 약물 처방 : 합병증 및 만성 약물 처방은 이론적으로 진단 테스트에 영향을 줄 수 있다. 진단받은 환자 중 62 %는 합병증이 없었으며 38 %는 하나 이상의 만성 질병을 가지고 있었다. 또한, 75 %의 환자는 만성 약물로 치료받지 않았으며 25 %는 하나 이상의 만성 약물로 치료되었다. 우리 환자 집단에서 가장 흔한 만성 질환은 고혈압, 고지혈증, 폐 질환 (COPD, 천식 등), 당뇨병 (대부분 2 형) 및 허혈성 심장 질환으로 서양에서 가장 흔한 만성 질병을 반영한다 (도 12a). 우리 환자 집단에 의해 사용된 만성 약물 분포는 보고된 만성 질병의 범위와 강하게 관련되어 있었다 (예: 합병증 환자의 29 %가 고지혈증을 앓고 있고 지질 강하제가 가장 많이 사용되는 약물이었다). 기타 자주 사용되는 약물에는 아스피린, 혈당 조절 약물 및 베타 차단제가 포함되었다 (도 12b).
환자 모집군 : 소아 환자 (18세 이하)는 소아과 응급실 (PED), 소아과 및 외과 부서로부터 모집하였고 성인 환자(18 세 초과)는 응급실 (ED), 내과 및 외과로부터 모집하였다. 소아용 ED는 가장 보편적인 모집 사이트 (39 %) 였고 다른 사이트들도 상대적으로 균형잡힌 모집 절차를 반영(17-20%)하여 견줄만 하였다. ED 환자와 입원 환자의 비율은 성인의 경우 ~1:1이었고 소아의 경우 ~2:1이었다 (도 13).
제외된 환자의 특성: 연구에 모집된 1002 명의 환자 중 110 명 (11 %)이 제외되었다 (일부 환자는 하나 이상의 제외 기준을 충족함). 가장 흔한 이유는 37.5 ℃ 이하의 발열을 가진 경우(n=54), 그 다음 증상 발현으로부터 의 시기가 12 일 (12 일)이 초과한 경우(n=26) 및 최근(14일) 감염성 질병을 가진 경우 (n=22)이었다. 다른 제외 이유는 활동성 악성 종양 (n=14)을 가진 경우 및 면역 저하 상태(예: 면역 억제 약물 치료로 인해; n=2)인 경우였다.
불확실한 환자의 특성 : 실험실 및 임상 정보의 엄격한 수집에도 불구하고 전문가 패널이 합동 기준 표준을 안정적으로 수립할 수 없기 때문에 총 98 명의 환자가 불확실한 것으로 정의되었다. 기준 표준이 없는 경우 이러한 환자의 시그니쳐 성능을 직접 검사할 수는 없지만 기준 표준을 가진 환자와 다른지 여부를 평가하기 위해 숙주-단백질 반응을 분석할 수 있다. 발명자들은 기준 표준이 있는 급성 감염 환자(n=653)와 기준 표준이 없는 급성 감염 환자(n=98)의 TRAIL, IP-10 및 CRP의 분포를 비교했다. 통계적으로 유의한 차이는 관찰되지 않았다 (Kolmogorov Smirnov 테스트; TRAIL, IP-10 및 CRP 각각 P = 0.20, 0.25, 0.46). 기준 표준이 있는 환자와 없는 환자 사이의 숙주-단백질 반응의 유사성은 본 방법의 접근방법이 임상 환경에서 불확실한 환자를 진단하는데 유용할 수 있음을 의미한다.
시그니쳐 성능은 상이한 환자 서브 그룹에 걸쳐 견고하게 유지된다 : 실시예 1에서, 본 발명자는 연령, 임상 증후군, 증상 발현으로부터의 시간, 최대 온도, 병원균 종, 합병증 및 AUC가 0.87에서 1.0 사이인 임상 사이트를 포함하는 다양한 환자 특성에 걸쳐 견고함을 유지함을 입증하였다 (도 4). 이 실시예에서 추가적인 환자 하위 그룹 전체의 시그니쳐 성능의 검토가 제공된다.
만성 약물 처방에 의한 계층화 : 실제 임상에서 환자는 흔하게 다양한 만성 약물 처방을 받고 있으며 이는 잠재적으로 시그니쳐를 구성하는 단백질 레벨에 영향을 줄 수 있다. 따라서 본 발명자들은 집단에서 가장 많이 사용되는 약물 (카테고리별)이 시그니쳐의 성능에 영향을 주는지 여부를 조사했다. 평가된 약물 그룹 중 어느 것도 시그니쳐의 정확성에 큰 변화를 일으키지 않았다 (표 5).
| 약물 분류 | AUC [95% CI] | 전체 환자, n | 박테리아성 환자, n | 바이러스성 환자, n | |
| Anti Hypertensive | 0.95 | [0.90, 1.00] | 50 | 43 | 7 |
| Anti platelets | 0.99 | [0.96, 1.00] | 54 | 48 | 6 |
| Anti-acid | 0.90 | [0.80, 1.00] | 42 | 35 | 7 |
| Antidepressants | 0.98 | [0.93, 1.00] | 29 | 25 | 4 |
| Beta Blocker | 0.95 | [0.88, 1.00] | 40 | 35 | 5 |
| Ca Channel Blocker | 0.94 | [0.86, 1.00] | 39 | 34 | 5 |
| Cholesterol / TG Lowering | 0.94 | [0.89, 1.00] | 64 | 53 | 11 |
| Diabetic | 0.87 | [0.74, 1.00] | 40 | 35 | 5 |
| Diuretics | 0.93 | [0.83, 1.00] | 30 | 25 | 5 |
| Hormonal | 0.98 | [0.93, 1.00] | 18 | 14 | 4 |
| Inhaled CS | 0.95 | [0.87, 0.99] | 26 | 18 | 8 |
| Prostate Hypertrophy | 0.94 | [0.84, 1.00] | 25 | 21 | 4 |
패혈증 기반 계층화 : 패혈증은 전신 염증성 상태 (전신성 염증 반응 증후군 (SIRS)이라고 함) 및 감염이 있거나 의심되는 것으로 나타나는 잠재적인 치명적 질환이다. 박테리아성 패혈증 환자는 조기 항생제 치료의 혜택을 받는다; 지연되거나 오진될 경우 심각하거나 치명적인 결과를 초래할 수 있다. 본 발명자들은 SIRS의 정의가 명확한 성인 환자에 초점을 맞추고 박테리아성 패혈증을 가진 성인 환자와 바이러스성 감염이 있는 성인 환자뿐만 아니라 박테리아성 패혈증을 가진 성인 환자와 바이러스성 패혈증 성인 환자를 구별하는 시그니쳐의 능력을 조사하였다.
박테리아성 패혈증을 가진 성인 환자는 American College of Chest Physicians and the Society of Critical Care Medicine에 따라 정의되었다. SIRS는 다음 중 두가지 이상에 해당하는 경우로 정의된다: (i) 체온이 <36 ℃ 또는 >38 ℃, (ii) 심장 박동 분당 >90 박자, (iii) 호흡률 분당 >20 회 또는 혈중 가스 PaCO2 <32 mm Hg (4.3 kPa), 및 (iv) WBC가 <4,000 세포/mm3 또는 >12,000 세포/mm3 또는 >10 % 밴드 형태. 이 시그니쳐가 박테리아성 패혈증을 가진 성인 환자와 바이러스성 패혈증 환자를 식별하는데 매우 높은 정확도를 가짐을 확인하였다(만장일치 집단[성인 박테리아성 패혈증, 성인 바이러스성 패혈증] 및 대다수 집단[성인 박테리아성 패혈증, 성인 바이러스성 패혈증]에 대해 AUC는 각각 0.97 및 0.93). 이 결과는 바이러스성 감염이 있는 성인 환자로부터 박테리아성 패혈증을 가진 성인 환자를 식별하는데 있어서 시그니쳐의 유용성을 입증한다.
| AUC [95% CI] | 전체 환자, n | 박테리아성 환자, n | 바이러스성 환자, n | ||
| 만장일치 집단 | 0.97 | [0.94, 1.00] | 114 | 93 | 21 |
| 대다수 집단 | 0.93 | [0.89, 0.97] | 147 | 112 | 35 |
박테리아성 대 비-박테리아성 환자 계층화 : 항생제 오용은 전형적으로 비 박테리아성 (바이러스성 또는 비감염성) 환자를 치료하거나 박테리아성 감염의 진단이 지연되거나 누락되어 이들 약물을 사용함에 기인한다.
따라서, 본 발명자들은 박테리아성 및 비-박테리아성 환자를 식별하기 위한 시그니쳐 성능을 추가로 조사하였다. 대부분의 대다수 집단은 leave-10 % out 교차 검증을 사용하여 평가되었으며, 0.94 ± 0.02의 AUC를 산출했다. 만장 일치 집단 (AUC 0.96 ± 0.02)를 평가할 때 및 한계 면역 반응이 있는 환자를 걸러 내고 향상된 결과가 나타났다(표 7).
| 모든 환자 | 한계 면역 반응 필터 | |||
| Accuracy measure | 만장일치 집단 | 대다수 집단 | 만장일치 집단 | 대다수 집단 |
| AUC | 0.96 (0.94, 0.98) |
0.94 (0.92, 0.96) |
0.96 (0.94, 0.98) |
0.95 (0.93, 0.97) |
| Total accuracy | 0.91 (0.89, 0.93) |
0.88 (0.85, 0.91) |
0.93 (0.91, 0.95) |
0.91 (0.89, 0.93) |
| Sensitivity | 0.88 (0.85, 0.91) |
0.87 (0.83, 0.91) |
0.92 (0.88, 0.95) |
0.91 (0.88, 0.95) |
| Specificity | 0.93 (0.91, 0.95) |
0.90 (0.87, 0.93) |
0.94 (0.91, 0.96) |
0.92 (0.89, 0.95) |
| LR+ | 12.6 (9, 18) |
8.7 (6, 12) |
15.3 (10, 23) |
11.4 (8, 16) |
| LR- | 0.13 (0.09, 0.18) |
0.14 (0.11, 0.19) |
0.08 (0.05, 0.13) |
0.1 (0.07, 0.14) |
| DOR | 97 (56, 168) |
60 (38, 94) |
180 (94, 344) |
116 (67, 200) |
상이한 온도에서의 단백질 안정성은 분석 성능에 영향을 미칠 수 있다 : 바이오마커의 유용성은 실제 임상 환경에서의 안정성에 달려있다 (예를 들어, 샘플이 분석물 측정 전에 실온에서 저장될 때의 분해율). 이를 해결하기 위해 우리는 4 명의 독립적인 개인의 혈청 샘플에서 TRAIL, CRP 및 IP-10의 안정성을 4 ℃ 및 25 ℃에서 24 시간 동안 조사했다. 각 혈장 샘플에서 100 μL의 분취량을 0.2 mL 튜브에 분주하고 0에서 24 시간 사이 4 ℃ 또는 25 ℃에서 유지하였다. 이어서, 분석물의 레벨을 측정하였다 (동일한 분석물에 대해 다른 시점에 동일한 플레이트 및 시약을 사용하여 측정하였다). 4 ℃ 및 25 ℃에서 상기 분석물의 반감기는 TRAIL, CRP 및 IP-10에 대해 72 시간 이상이었다 (도 15a-15c). 실제 임상 환경에서 샘플을 실온에서 보관할 경우 샘플을 얻은 후 TRAIL, IP-10 및 CRP의 농도는 약 24 이내에 측정해야 한다. 바람직하게는 이들을 5 시간, 4 시간, 3 시간, 2 시간, 1 시간 이내에 또는 샘플을 얻은 직후에 측정해야 한다. 또는 샘플을 10 ℃ 이하의 온도에서 보관해야 하며 샘플을 얻은 후 TRAIL을 24 회 이상 측정 할 수 있다.
세 가지 단백질 조합은 모든 개별 및 단백질 쌍보다 우수하다 : 세 가지 단백질의 조합은 박테리아성 대 바이러스성 및 감염성 대 비-감염성 환자를 식별하기 위해 개별 및 단백질 쌍의 성능을 능가한다.
| 단백질 #1 | 단백질#2 | 단백질 #3 | AUC |
| TRAIL | - | - | 0.89 |
| CRP | - | - | 0.88 |
| IP-10 | - | - | 0.66 |
| TRAIL | CRP | - | 0.95 |
| CRP | IP-10 | - | 0.93 |
| TRAIL | IP-10 | - | 0.90 |
| TRAIL | CRP | IP-10 | 0.96 |
| 단백질 #1 | 단백질 #2 | 단백질 #3 | AUC |
| TRAIL | - | - | 0.60 |
| CRP | - | - | 0.87 |
| IP-10 | - | - | 0.89 |
| TRAIL | CRP | - | 0.90 |
| CRP | IP-10 | - | 0.95 |
| TRAIL | IP10 | - | 0.89 |
| TRAIL | CRP | IP-10 | 0.96 |
박테리아성 감염의 유병률에 대한 함수로써 성능 분석: 박테리아성 및 바이러스성 감염의 유병률은 환경 의존적이다. 예를 들어, 겨울에는 외래 환자의 소아과 의사가 여름 동안 병원 내과의 의사보다 훨씬 더 많은 바이러스성 감염을 경험할 것으로 예상된다. 주목할만하게, AUC, 민감도 및 특이성과 같은 진단 정확도의 일부 척도는 근복적으로 유병률에 불변하지만, PPV 및 NPV와 같은 다른 정확성 척도는 유병율에 좌우된다. 이 섹션에서는 박테리아성 및 바이러스성 감염에 대한 상이한 유병률을 가진 임상적 환경에서 PPV 및 NPV 측면에서 예상되는 시그니쳐 성능을 검토하였다.
이 분석을 위한 기초로서 만장일치 (박테리아성, 바이러스성) 및 대다수 (박테리아성, 바이러스성) 집단을 사용하여 얻은 상기 시그니쳐 정확성 척도들을 사용하였다. 만장일치 집단에서 박테리아성 감염의 유병률은 93% ± 3% PPV 및 93% ± 3% 및 NPV를 산출하는 51.7%였다. 대다수 집단에서 박테리아성 감염의 유병률은 89 % ± 3 %의 PPV와 92 % ± 3 %의 NPV를 산출하는 48.7%였다.
측정된 민감도와 특이성은 박테리아성 감염의 유병률에 대한 함수로써 시그니쳐 PPV와 NPV에서 예상되는 변화를 계산하는데 사용되었다 (도 14a-14b).
각기 다른 임상적 환경 및 외삽된 시그니쳐 PPV 및 NPV의 실시예를 표 10에 나타내었다.
| 환경 | 연령 | 박테리아성 감염 유병률* | PPV | NPV |
| 외래환자 | 소아 | 20% | 76% | 98% |
| 외래환자 | 성인 | 35% | 85% | 97% |
| 입원환자 | 소아 | 50% | 93% | 94% |
| 입원환자 | 성인 | 80% | 98% | 78% |
* 평균 연간 유병률. 박테리아성 감염 유병률 척도는 Bacterial etiology chapter, Part 7 of Harrison’s Internal Medicine 17th Edition에 보고된 데이터에 기초한다.
상기 시그니쳐는 박테리아성 대 바이러스성 감염을 진단하기 위한 표준 실험실 및 임상 파라미터를 능가한다 : 감염원의 식별 진단을 돕기 위해 임상 실험에서 일상적으로 사용되는 일부 표준 실험실 및 임상 파라미터를 대다수 집단 (박테리아성, 바이러스성, 비 감염성, n = 765)에서 평가하였다. 평가된 파라미터는 ANC, 호중구 %, 림프구 %, WBC 및 최대 온도를 포함하였다. 이러한 파라미터의 확립된 임상적 역할에 따라 박테리아성 및 바이러스성 환자 간에 통계적으로 유의한 차이를 관찰하였다 (도 15A-E). 예를 들어 박테리아성 환자의 경우 ANC (P <10-24) 및 WBC (P <10- 10)의 레벨이 증가하는 반면 바이러스성 환자의 경우 림프구 % 가 더 높았다 (P <10-31). 상기 시그니쳐는 개별 (P<10-18) 및 그 조합 (P<10-15) 중 어느 것보다 훨씬 더 정확하였다 (도 3a 참조).
상기 시그니쳐는 잘 확립된 면역학적 역할을 가진 단백질 바이오마커를 능가한다. 상기 시그니쳐는 스크리닝 단계에서 평가된 모든 임상 파라미터와 600 개의 단백질보다 우수하다 (도 3a-3b 참조). 다음 절에서는 임상적 환경에서 일상적으로 사용되거나 면역학적 역할을 하는 선택된 단백질에 대한 상기 시그니쳐를 비교하였다.
중환자에서 SIRS의 다른 비 감염 원인으로부터 패혈증을 구별하기 위해 가장 널리 사용되고 유용한 단백질 바이오마커 중 하나는 프로 칼시토닌 (procalcitonin, PCT)이다. PCT가 국소 박테리아성 및 바이러스성 감염을 구별하는데 사용될 수 있는지 여부는 명확하지 않다. 이를 테스트하기 위해 만장일치 (박테리아성, 바이러스성) 집단 (n박테리아성=39, n바이러스성=37)으로부터 무작위로 선택된 76명의 환자들 및 대다수 (박테리아성, 바이러스성) 집단 (n박테리아성=51, n바이러스성=50)으로부터 무작위로 선택된 101명의 환자들에서 PCT 농도를 측정하였고 PCT 수준에 따른 진단 정확도를 시그니쳐의 정확도와 비교하였다. PCT 정확도를 임상적 환경 (0.1 ng/mL, 0.25 ng/mL, 0.5 ng/mL 및 1 ng/mL)에서 일상적으로 적용되는 표준 cutoffs를 사용하여 계산하였다. 69 %의 최대 PCT 민감도는 0.1mg / mL의 컷오프에서 달성되었으며 62 %의 특이도를 나타냈다 (만장일치 [박테리아성, 바이러스성] 집단의 경우). 동일한 집단의 경우, 상기 시그니쳐는 상당히 높은 94 %의 민감도 (P <0.001) 및 93 %의 특이성 (P <0.001)를 보였다 (도 16a). 대다수 (박테리아성, 바이러스성) 집단의 환자를 사용한 비교에서도 비슷한 결과가 나타났다 (도 16b).
전반적으로 중환자의 패혈증 진단에 대한 높은 진단 및 예후값에도 불구하고 우리의 결과는 PCT가 국소 감염 (박테리아성 대 바이러스성) 환자를 구별하는데 덜 정확함을 보여준다.
임상 환경에서 사용되는 또 다른 단백질 바이오마커는 감염 및 기타 염증성 질환에서 증가하는 급성 반응 단백질인 C-반응성 단백질 (CRP)이다. CRP의 성능은 만장 일치 (박테리아성, 바이러스성) 및 대다수 (박테리아성, 바이러스성) 집단을 사용하는 시그니쳐의 성능과 비교되었다. CRP 정확도는 임상 환경에 적용된 몇 가지 표준 컷오프 (cutoffs)를 사용하여 결정되었다. 최대 CRP 민감도는 20 mg/mL cutoff에서 달성되어 60 %의 특이도를 보였다 (만장일치(박테리아성, 바이러스성) 집단의 경우)(도 17a). 동일한 집단에서 상기 시그니쳐는 비슷한 민감도 (94 %) 및 특이성 (93 %, P <10-9)을 나타냈다. 대다수 (박테리아성, 바이러스성) 집단을 사용한 경우 유사한 결과가 관찰되었다(도 17b). 전반적으로, 상기 시그니쳐는 20 mg/L 컷오프의 CRP와 유사한 민감성을 갖지만 박테리아성와 바이러스성 환자를 식별하기 위한 상당히 높은 특이성을 가진다.
다음으로, 감염에 대한 숙주 반응에서 잘 확립된 역할을 하는 단백질 바이오마커의 차별적 반응을 조사하였다 (표 11 및 도 18a-18h). 각각의 바이오마커는 최소 43 명의 환자 (약 절반의 박테리아성 및 절반의 바이러스성)에서 테스트되었으며, 유망한 결과가 나온 경우 추가 환자 (최대 150 명)에 대해 추가로 테스트하였다.
| 단백질 바이오마커 | 요약 | 환자 수 |
| CD11a | CD11a는 인테그린 림프구의 일부로서 모든 백혈구에 의해 발현된다 기능 관련 항원-1 (LFA-1). LFA-1은 리간드인 ICAM 1-3 (세포간 부착 분자 1-3)과의 상호 작용을 통해 백혈구 세포 간 부착에 중심적인 역할을 한다. CD11a는 또한 림프구 공동 자극 신호전달(lymphocyte co-stimulatory signaling)에 이용된다. |
120 |
| CD11C | CD11C는 인테그린 α X 사슬 단백질이며 염증 반응 동안 세포-세포 상호 작용을 매개한다 |
79 |
| CD80 | CD80은 T- 림프구 활성화에 필수적인 보조 자극 신호에 관여하는 막 수용체이다. CD80에 대한 CD28 또는 CTLA-4의 결합은 T 세포 증식 및 사이토카인 생산을 유도한다. |
82 |
| HLA-A,B,C | 이들은 β2-마이크로글로불린과 관련된 MHC 클래스 I 항원이며 모든 인간의 유핵 세포에 의해 발현된다. HLA-A, B, C는 세포 매개 면역 반응과 종양 감시 센터이다. | 65 |
| IFN-γ | IFN-γ는 수용성 사이토카인이다. IFN-γ는 바이러스성 및 세포 내 박테이라 감염 및 종양 조절에 대한 선천적 적응 면역에 참여한다. |
49 |
| IL-1a | IL-1a는 IL-1 사이토카인의 구성원이다. IL-1a는 다양한 면역 반응, 염증 과정 및 조혈에 관여하는 다면발현 사이토카인이다. IL-1a는 프로테아제로서 단핵 세포 및 대 식세포에 의해 생산되며, 단백질 분해 처리되고 세포 손상에 반응하여 방출되어 세포 사멸을 유도한다. |
43 |
| IL-2 | IL-2는 항원 자극 또는 분열 촉진 자극에 반응하여 T 세포에 의해 생성된다. IL-2는 면역 반응 조절에 중요한 T 세포 증식 및 기타 활동에 필요하다. | 49 |
| IL-6 | IL-6는 B 세포의 염증 및 성숙에 작용하는 사이토카인이다. IL-6는 자가 면역 질환이나 감염자에게 열을 일으킬 수 있는 내인성 피로겐(pyrogen)이다. | 43 |
| IL-8 | IL-8은 CXC 케모카인 계통의 구성원이며 염증 반응의 주요 매개자 중 하나로서 기능한다. | 43 |
| IL-9 | IL-9는 다양한 조혈 세포의 조절 인자로서 작용하는 사이토카인이다. IL-9는 헬퍼 T 세포의 IL-2 독립적 및 IL-4 독립적인 성장을 보조한다. |
43 |
| IL-10 | IL-10은 주로 단핵구에 의해 생성되는 사이토카인이며, 보다 적게는 림프구에 의해 생성된다. IL-10은 면역 조절 및 염증에 있어서 다면 발현 효과를 갖는다. | 48 |
| IL-15 | IL-15는 T-림프구의 증식을 자극하는 사이토카인이다. | 49 |
| IL-16 | IL-16은 화학 유인 물질, T 세포 활성화의 조절 인자 및 HIV 복제 억제제로서 기능한다 | 49 |
| sTNFRSF1A | sTNFRSF1A는 항 바이러스성 상태 및 산성 스핑고미엘리나제 (sphingomyelinase)의 활성화를 포함하여 비-세포 파괴 TNF 효과의 유도에 기여하는 TNFSF2/TNF-α 및 호모-트리머릭(homo-trimeric) TNFSF1/림포톡신-α에 대한 수용체이다. | 54 |
| TNF-α | TNF-α는 주로 대식세포에 의해 분비되는 사이토카인이다. TNF-α는 특정 종양 세포주의 세포 사멸을 유도할 수 있다. 직접적으로 또는 IL-1 분비의 자극에 의해 발열을 일으키는 강력한 발열 촉진제이다. | 43 |
| TNF-β | TNF-β는 염증 반응 및 면역 반응의 유력한 매개자이다. 이것은 활성화된 T 및 B 림프구에 의해 생성되며 세포 증식, 분화, 세포 사멸, 지질 대사, 응고 및 신경 전달을 비롯한 다양한 생물학적 과정의 조절에 관여한다. | 43 |
| TREM | TREM은 호중구와 단핵구에 존재하는 전염증 촉진제이다. | 150 |
이들 바이오마커는 임상 환경에서 잘 확립된 컷오프가 없으므로 비교를 위한 기초로 AUC를 사용했다 (도 3b). 가장 유용한 바이오마커는 TREM (AUC 0.68 ± 0.09)이다. TREM의 정확도는 시그니쳐의 정확도보다 현저히 낮았다 (두 개의 AUC를 비교할 때 P <10-9, 도 3b). 이 결과는 감염에 대한 숙주 반응에 단백질이 단순히 참여한다고 해서 반드시 진단에 유용함을 의미하지는 않는다는 것을 보여준다. 예를 들어, IFN-γ는 바이러스성 및 세포 내 박테리아성에 대한 면역 반응에서 확립된 역할을 하지만 그것의 짧은 반감기 (<20 시간)는 진단 유용성을 제한한다 (혈중 농도가 감염 발생으로부터의 기간에 매우 의존적이기 때문).
실시예
3
삼중 분류기(
Trinary
classifier)는 이중 분류기(binary classifier)보다 우수하다.
이중 모델에서 모든 샘플을 '박테리아성' 또는 '비 박테리아성'('바이러스성 성' 및 '비 감염성'으로 그룹화됨)로 분류함으로ㅆ 분류기를 트레이닝한다. 삼중 모델에서, 분류기는 세 가지 클래스, '박테리아성', '바이러스성' 및 '비 감염성'을 구별한다. 그 다음 '비 박테리아성'의 확률을 제공하기 위해 바이러스성 및 비-감염성의 확률이 그룹화된다. 이것은 본 발명의 데이터에서 증명되었다.
상기 분류기는 둘 다 대다수 및 만장일치 집단에 대해 leave 10%-out cross-validation을 사용하여 평가되었다.
결과
대다수 집단에서 이중 분류기를 실행하면 다음의 표 12에 요약된 결과가 산출된다:
|
참조 클래스
|
||
|
바이러스성 및
비 감염성
(V+ NI ) |
박테리아성
(B) |
|
| V+ NI | 411 | 63 |
| B | 35 | 256 |
대다수 집단에 대한 분류기의 민감도는 80.3 %이고 특이성은 92.2 %이다.
동일한 데이터 세트에서 다항식 기반 분류기를 실행하면 표 13에 요약된 다음 결과가 산출된다.
|
참조 클래스
|
||
| (V+ NI ) | (B) | |
| V+ NI | 417 | 54 |
| B | 29 | 265 |
이 분류기는 민감도와 특이성 측면에서 이전 분류기보다 우수하다는 것을 알 수 있다. 민감도는 83.1 %로 특이성은 93.5 %로 향상되었다.
만장일치 집단에서 이중 분류기를 실행하면 표 14에 요약된 결과가 산출된다.
|
참조 클래스
|
||
| (V+ NI ) | (B) | |
| V+ NI | 358 | 39 |
| B | 25 | 217 |
만장 일치 집단에 대한 분류기의 민감도는 84.8 %이며 특이성은 93.5 %이다.
동일한 데이터 세트에서 다항식 기반 분류기를 실행하면 표 15에 요약된 결과가 산출된다.
|
참조 클래스
|
||
| (V+ NI ) | (B) | |
| V+ NI | 364 | 38 |
| B | 19 | 218 |
이 분류기는 민감도와 특이성 측면에서 이전 분류기보다 우수하다. 민감도는 85.2 %로 특이성은 95.0 %로 향상되었다.
요약하면, 삼중 분류기는 테스트된 두 데이터 세트에 대한 민감도 및 특이성 측면에서 이중 분류기보다 성능이 우수하다.
실시예
4
분석 정확도가 떨어지더라도
시그니쳐의
임상적 정확성은
견고하다
종종 다른 측정 장치, 특히 현장 진료(point-of-care)에서 유용한 측정 장치가 증가된 CV를 보여주기 때문에(즉, 감소된 분석 정확도) 임상적 정확성이 단백질 측정의 CV (표준/평균)의 증가에 어떻게 영향을 받는지 평가하는 것이 중요하다.
본 발명자들은 TRAIL 및 CRP 모두에 대한 CV의 증가 함수로서 박테리아성 및 바이러스성 감염을 구별하기 위해 시그니쳐의 AUC의 변화를 조사하였다. 이것은 만장일치 집단의 최초 환자 데이터를 취하고 몬테카를로 시뮬레이션(monte-carlo simulations)을 사용하여 CV의 증가를 시뮬레이션함으로써 수행되었다 (도 19A-B). 특히, TRAIL및 CRP CV의 각 조합에 대해 100 개의 시뮬레이션 측정이 각 환자에게 할당되었고 각 경우에 AUC가 다시 계산되었다. CV 조합당 평균 AUC가 도시된다. 시그니쳐 임상적 정확성 (AUC 기준)은 기술적 CV의 증가에 견고하다는 것을 알 수 있다. 예를 들어, ELISA CV를 0, 0.24 및 0.4만큼 증가시키면 AUC가 0.96, 0.95 및 0.94로 각각 감소한다. IP-10의 CV를 증가시킬 때 및 대다수 집단에 대한 시뮬레이션을 반복할 때 유사한 결과가 얻어진다.
이 결과는 서로를 보상하는 다중 바이오마커의 사용으로 설명될 수 있다. 이 놀라운 발견은 임상적 정확성을 잃지 않으면서 감소된 분석 민감도 (예 : 자동화된 면역 측정법 또는 ELISA와 비교)를 나타내는 저렴하고 신속한 기술(예 POC 기술)로 단백질 측정을 수행할 수 있는 길을 열어주기 때문에 유용하다.
실시예
5
서로 다른 ELISA 프로토콜을 TRAIL 및 IP-10 측정에 적용할 수 있고, 이는 신호 대 잡음비가 달라질 수 있어 결과적으로 다른 농도로 측정된다. 보다 구체적으로, 바이오마커의 전반적인 경향은 프로토콜 (예: 바이러스성 감염에서 TRAIL 증가 및 박테리아성 감염에서 TRAIL 감소)과 상관없이 보존되지만, 측정 규모는 프로토콜에 좌우된다. 다음 하위 절에서는 IP-10 및 TRAIL의 측정 농도를 다르게 하는 프로토콜의 예를 설명한다.
ELISA를 이용한 가용성 IP -10 및 TRAIL의 측정 프로토콜 no. 1: 인간 혈장 및 혈청 샘플에서 가용성 IP-10 및 TRAIL의 농도를 결정하기 위해 표준 샌드위치 ELISA(standard Sandwich Enzyme-linked immunosorbent assay)를 사용하였다. 간단히, 96-웰 플레이트의 웰(well)을 TRAIL 및 IP-10에 특이적인 포집-항체로 코팅하고 코팅 버퍼 (예 : 1xPBS)에서 희석한 후 4 ℃에서 밤새 인큐베이션하였다. 웰을 세척 버퍼 (예: 0.2 % Tween-20을 포함한 1xPBS)으로 2 회 세척한 후 실온에서 최소 2 시간 동안 또는 밤새 밤새 4 °C에서 단백질을 함유한 차단 버퍼 (예: 0.2 % Tween-20 및 5 % 탈지유가 포함된 1xPBS)로 차단하였다. 그 다음, 웰을 세척 버퍼로 2 회 세척하였다. 단백질 표준 및 혈장 또는 혈청 샘플을 실온에서 2 시간 동안 인큐베이션하였다. 이어서, 웰을 세척 버퍼로 3 회 세척한 후 실온에서 차단 버퍼에서 2 시간 동안 희석된 TRAIL 및 IP-10에 특이적인 HRP 접합 검출 항체와 함께 인큐베이션하였다.
웰을 세척 버퍼로 4 회 세척하고 HRP 기질 (예 : TMB; 3,3',5,5'- 테트라메틸벤지딘)을 함유하는 반응 용액과 함께 인큐베이션하였다. 적절하게 발색시킨 후, 정지 용액을 각 웰에 첨가하였다. 450nm에서의 HRP 반응 생성물의 흡광도는 표준 분광 광도계를 사용하여 측정하였다. 이 프로토콜은 각각 5 (TRAIL) 및 4.75 (IP10) 시간이 걸렸으며 본 명세서에서는 느린 프로토콜이라고 지칭된다.
ELISA를 이용한 가용성
IP
-10 및 TRAIL의
측정 프로토콜
no. 2:
분석 시간을 줄이면 임상적 유용성이 증가한다. 프로토콜 실행 시간을 더 줄이기 위해 TRAIL 및 IP10 측정을 위한 프로토콜을 최적화했으며 100분 미만으로 단축하였다. 빠른 프로토콜은 다음과 같이 수행되었다:
분석용 희석액 50㎕와 표준 용액 50㎕를 샘플 또는 대조군에 웰 당 첨가하였다. 상기 반응물을 550 rpm으로 설정된 수평 궤도 마이크로 플레이트 쉐이커 (3mm 궤도)에서 30분 동안 실온에서 인큐베이션하였다. 이어서, 각 웰을 흡인하고 세척 버퍼을 사용하여 4회 세척하였다. 그 다음, 200㎕의 접합체(Conjugate)를 각 웰에 첨가하고, 반응물을 쉐이커에서 상온에서 45분 동안 인큐베이션하였다. 웰을 세척 버퍼로 4회 세척하고 HRP 기질 (예 : TMB; 3,3',5,5'- 테트라메틸벤지딘)을 함유하는 반응 용액과 함께 인큐베이션하였다. 10-25분 후, 정지 용액을 각 웰에 첨가하였다. 450nm에서의 HRP 반응 생성물의 흡광도는 표준 분광 광도계를 사용하여 측정하였다. 이 프로토콜은 각각 99 (TRAIL) 및 85 (IP-10) 분이 걸렸으며 본 명세서에서는 빠른 프로토콜이라고 지칭된다.
느린 프로토콜과 빠른 프로토콜 측정을 TRAIL에 대한 357 샘플과 IP-10에 대한 189 샘플을 사용하여 비교하였고 높은 상관 관계가 있는 결과를 나타냈다 (도 30A-B).
참고로, 빠른 프로토콜을 사용하여 얻은 평균 TRAIL 농도는 느린 프로토콜 농도를 사용하여 얻은 것보다 약 70 % 묽었다. 서로 다른 프로토콜을 통해 측정된 단백질 농도의 이러한 변화는 종종 발생하며, 두 프로토콜의 측정치를 상호 관련시키고 변환 기능을 계산함으로써 보완할 수 있다. 예를 들어, 변환 함수 y_slow = 0.709 x y_rapid -3e-12는 빠른 프로토콜과 느린 프로토콜의 농도를 변환하는 데 사용될 수 있다. 이 변환은 TRAIL의 정확성을 유지한다. 또한, 통상의 기술자는 정확성을 유지하는 다른 변환 함수 및 프로토콜을 개발할 수 있다. 요약하면, TRAIL의 행동은 계산된 농도의 변화에도 불구하고 두 프로토콜 (바이러스성에서 가장 높고, 비 감염성에서 낮고, 박테리아성에서 가장 낮음)에서 동일하게 유지된다.
다른 프로토콜 및 집단은 다른 모델 계수를
이끈다
느린 프로토콜로 IP-10 및 TRAIL을 측정할 때 대다수 환자 집단에서 생성된 다항 로지스틱 모델 계수의 예가 표 16에 보여진다 :
| 제 1좌표 δ0 (바이러스성) |
제 2
좌표
δ 1 (박테리아성) |
|
| Const | a 0 =-1.7331 ± 0.62936 | b 0 =-1.5389 ± 0.75676 |
| CRP (mg/ml) | a 1 = 0.0514 ± 0.014896 | b 1 = 0.0851 ± 0.015288 |
| IP10 ( pg /ml) | a 2 = 0.0049 ± 0.001372 | b 2 = 0.0046 ± 0.001372 |
| TRAIL ( pg /ml) | a 3 = 0.0048 ± 0.005096 | b 3 = -0.0155 ± 0.007056 |
느린 프로토콜로 IP-10 및 TRAIL을 측정할 때 만장일치 환자 집단에서 생성된 다항 로지스틱 모델 계수의 예가 표 17에 보여진다.
| 제 1좌표 δ0 (바이러스성) |
제 2
좌표
δ 1 (박테리아성) |
|
| Const | a 0 =-2.6866 ± 0.75048 | b 0 =2.6091±0.9357 |
| CRP (mg/ml) | a 1 = 0.0499 ± 0.016464 | b 1 = 0.0866 ± 0.016856 |
| IP10 ( pg /ml) | a 2 = 0.0059 ± 0.001568 | b 2 = 0.0052 ± 0.001568 |
| TRAIL ( pg /ml) | a 3 = 0.0084 ± 0.005684 | b 3 = -0.0115 ± 0.008232 |
환자 집단 내의 하위 그룹의 빈도가 일반 모집단에서 예상되는 빈도로부터 벗어나기 때문에, 계수 조정을 위한 표준 기술을 사용하여 미리 결정된 사전 확률을 반영하도록 모델 계수를 조정할 수 있다 (예를 들어, G. King and L Zeng, Statistics in Medicine 2002 참조). 예를 들어, 다음 예는 45 % 박테리아성, 45 % 바이러스성 및 10 % 비 감염성의 사전 확률을 반영하여 느린 프로토콜로 IP-10 및 TRAIL을 측정할 때 대다수 환자 집단에서 생성된 다항 로지스틱 모델 계수를 보여준다.
사전 조정 후 모델 계수 (대다수 집단에 대해 측정된)는 표 18에 요약되어 있다:
| 제 1 좌표 δ0 (바이러스성) |
제 2
좌표
δ 1 (박테리아성) |
|
| Const | a 0 =-1.4151 0.62936 | b 0 =-1.1302 0.75676 |
| CRP (mg/ml) | a 1 = 0.0514 0.014896 | b 1 = 0.0851 0.015288 |
| IP10 ( pg /ml) | a 2 = 0.0049 0.001372 | b 2 = 0.0046 0.001372 |
| TRAIL ( pg /ml) | a 3 = 0.0048 0.005096 | b 3 = -0.0155 0.007056 |
사전 조정 후 모델 계수 (만장일치 집단에 대해 측정된)는 표 19에 요약되어 있다.
| 제 1 좌표 δ0 (바이러스성) |
제 2
좌표
δ 1 (박테리아성) |
|
| Const | a 0 =-2.083 0.75048 | b 0 =-1.7833 0.9357 |
| CRP (mg/ml) | a 1 = 0.0499 0.016464 | b 1 = 0.0866 0.016856 |
| IP10 ( pg /ml) | a 2 = 0.0059 0.001568 | b 2 = 0.0052 0.001568 |
| TRAIL ( pg /ml) | a 3 = 0.0084 0.005684 | b 3 = -0.0115 0.008232 |
유의할 점은, 통상의 기술자에게 명백한 바와 같이, 유사한 결과를 생성하기 위해 계수의 다른 조합이 선택될 수 있다는 것이다. 측정된 단백질 농도에 영향을 미치는 단백질을 측정하기 위한 다른 프로토콜은 다른 모델 계수를 산출한다. 예를 들어, TRAIL 측정을 위한 빠른 프로토콜은 계산된 농도를 느린 프로토콜에서 계산된 농도의 약 70 %로 줄인다. 따라서 이것을 조정하는 한 가지 방법은 이 변화를 설명하기 위해 TRAIL의 모델 계수를 변경하는 것이다. 또 다른 방법은 TRAIL의 빠른 프로토콜 측정을 70 %로 나누고 느린 프로토콜용으로 개발된 상기 언급한 모델에 연결하는 것이다.
모델 정확도 및 교정 (즉, 특정 감염의 예상 위험 및 관찰된 위험 사이의 적합성)을 향상시키기 위해 단백질 측정에 로그 변환을 사용하는 것이 종종 바람직하다.
TRAIL과 IP-10의 로그 변환 모델의 예가 표 20에 묘사되어 있다 (모델은 만장일치 집단에서 측정되었다) :
| 제 1좌표 δ0 (바이러스성) |
제 2
좌표
δ 1 (박테리아성) |
|
| Const | a 0 =-14.8487 ± 3.3839 | b 0 =-5.9471 ± 3.3391 |
| CRP (mg/ml) | a 1 = 0.0437 ± 0.017052 | b 1 = 0.0833 ± 0.016856 |
| IP10 ( pg /ml) | a 2 = 2.0148 ± 0.4408 | b 2 = 1.3868 ± 0.48608 |
| TRAIL ( pg /ml) | a 3 = 0.8946 ± 0.61348 | b 3 = -0.788 ± 0.60505 |
실시예
6
초곡면
파라미터화
(Hypersurface
parameterization
)
Given the concentrations of CRP [C], TRAIL [T] 및 IP-10 [P]의 농도가 주어지면, 다음과 같이 정의한다:
δ0 = -1.299 + 0.0605 x [C] + 0.0053 x [P] + 0.0088 x [T]
δ1 = -0.378 + 0.0875 x [C] + 0.0050 x [P] - 0.0201 x [T]
그 다음 상기 확률은 다음과 같이 계산될 수 있다:
우리는 [C], [T], [P] 공간에서 초곡면을 다음과 같이 정의한다:
이것은 박테리아성 및 비-박테리아성 환자를 구별하는데 사용된다. 하나의 바람직한 실시예에서, 환자의 [C], [T], [P] 값이 주어지면 이면 환자는 박테리아성로 분류되고, 그렇지 않으면 비-박테리아성로 분류된다.
박테리아성 감염 및 비-박테리아성 감염을 구별하는데 사용할 수 있는 모든 초평면은 다음 두 개의 초곡면에 존재하는 것으로 정의한다:
ε1는 0 및 1사이의 임의의 수일 수 있다. 일부 바람직한 실시예에서 ε1는 0.5, 0.4, 0.3, 0.2 또는 0.1보다 작다.
ε0는 0 및 ω사이의 임의의 수일 수 있다. 일부 바람작힌 실시예에서 ε0는 0.5, 0.4, 0.3, 0.2 또는 0.1보다 작다.
표면의 그림 예는 실시예 7에서 제공된다.
실시예
7
분류의 그래픽 표현
도 20은 박테리아성 ('+'), 바이러스성 ('O') 및 비 감염성 ('^') 환자의 3 차원 시각화이다. 다른 환자 유형은 CRP (μg/ml), TRAIL 및 IP-10 (pg/ml) 농도 맵에서 구별되는 영역으로 매핑된다.
예를 들어, 다항 로지스틱 회귀를 사용하여 확률면을 생성하였다. 표면의 등고선은 TRAIL (y-축), CRP (x-축) 및 IP-10 농도에 대한 함수로서 도 21a-28c에 나타내었다. 도 21a, 도 22a, 도 23a, 도 24a, 도 25a, 도 26a, 도 27a, 도 28a는 바이러스성 감염 확률을 도시한다. 도 21b, 도 22b, 도 23b, 도 24b, 도 25b, 도 26b, 도 27b 및 도 28b는 박테리아성 또는 혼합 감염성의 확률을 나타낸다. 도 21c, 도 22c, 도 23c, 도 24c, 도 25c, 도 26c, 도 27c 및 도 28c는 비 감염성 또는 건강한 자의 확률을 나타낸다. 도 21a 내지 도 21c는 0 내지 100 범위의 IP10pg에 대응한다. 도 22a 내지 도 22c는 100 내지 200 범위의 IP10pg에 대응한다. 도 23a 내지 도 23c는 200 내지 300 범위의 IP10pg에 대응한다. 도 24a 내지 도 24c는 300 내지 400 범위의 IP10pg에 대응한다. 도 25a 내지 도 25c는 400 내지 500의 IP10pg에 해당한다. 도 26a 내지 도 26c는 500 내지 1000 범위의 IP10pg에 대응한다. 도 27a 내지 도 27c는 1000 내지 2000 범위의 IP10pg에 해당하며, 도 28a 내지 도 28c는 IP10pg이 2000 이상인 것에 대응한다.
박테리아성 또는 혼합된 감염성 환자는 '+', 바이러스성 감염은 'O' 및 비 감염성 또는 건강한 환자는 '^'로 표시된다. 낮은 레벨의 IP-10은 비 감염성 질병, 더 높은 레벨은 박테리아성 및 가장 높은 레벨은 바이러스성 감염과 관련이 있다. 낮은 레벨의 TRAIL은 박테리아성 감염, 높은 레벨은 비 감염성 및 건강 및 가장 높은 레벨은 바이러스성 감염과 관련이 있다. 낮은 레벨의 CRP는 비 감염성 질병과 건강한 대상체, 높은 레벨은 바이러스성 감염 및 가장 높은 레벨은 박테리아성 감염과 관련이 있다. 3 개 단백질의 조합은 개별 단백질 또는 쌍의 단백질보다 그 진단 성능이 우수한 확률 함수를 생성한다.
도 35a-d는 박테리아성 (도 35a), 바이러스성 (도 35b), 비 박테리아성 (도 35c) 및 비-감염성 (도 35d) 병인의 확률을 좌표 δ0 및 δ1에 대한 함수로 나타낸 등고선도이다. 상기 확률값의 범위는 0% (검정) 내지 100% (흰색)이다.
실시예
8
발현값
측정을 위한 예시적 프로토콜
일반적으로, 제한없이 TRAIL의 발현값은 ELISA 또는 자동 면역 분석법을 사용하여 측정할 수 있다; IP-10의 발현값은 ELISA 분석을 사용하여 측정될 수 있다; CRP의 발현값은 ELISA 또는 자동 면역 분석법을 사용하여 측정할 수 있다. CRP의 발현값은 포스포릴콜린에 대한 칼슘 의존성 결합에 기초한 기능 분석법을 사용하여 측정할 수 있다.
프로토콜 A:
TRAIL
발현값
측정에 적합한 프로토콜
(a)
샘플 내 존재하는 TRAIL을 항체를 사용하여 고체 지지체에 고정시키는 단계;
(b)
고정된 TRAIL을 TRAIL에 특이적으로 결합하는 제 2 항체와 접촉시키는 단계; 및
(c)
고정된 TRAIL에 결합한 항체의 양을 정량하는 단계.
IP
-10
발현값
측정에 적합한 프로토콜
(a)
샘플 내 존재하는 IP-10을 포집 항체를 사용하여 고체 지지체에 고정시키는 단계;
(b)
고정된 IP-10을 IP-10에 특이적으로 결합하는 제 2 항체와 접촉시키는 단계; 및
(c)
고정된 IP-10에 결합한 항체의 양을 정량하는 단계.
CRP
발현값
측정에 적합한 프로토콜
(a)
샘플 내 존재하는 CRP를 포집 항체를 사용하여 고체 지지체에 고정시키는 단계;
(b)
고정된 CRP를 CRP에 특이적으로 결합하는 제 2 항체와 접촉시키는 단계; 및
(c)
고정된 CRP에 결합한 항체의 양을 정량하는 단계.
프로토콜 B:
TRAIL
발현값
측정에 적합한 프로토콜
(a) 샘플을 TRAIL에 특이적으로 결합하는 제1 항체와 인큐베이션하는 단계로, 상기 제 1항체는 고체상으로 고정된다;
(b) 세척하는 단계;
(c) TRAIL에 특이적으로 결합하는 제 2 항체를 첨가하는 단계로, 상기 제 2 항체는 효소와 컨쥬게이트된다; 및
(d) 효소 기질을 첨가하여 고정된 샘플에 결합하는 항체의 양을 정량하는 단계.
IP
-10
발현값
측정에 적합한 프로토콜
(a) 샘플을 IP-10에 특이적으로 결합하는 제1 항체와 인큐베이션하는 단계로, 상기 제 1항체는 고체상으로 고정된다;
(b) 세척하는 단계;
(c) IP-10에 특이적으로 결합하는 제 2 항체를 첨가하는 단계로, 상기 제 2 항체는 효소와 컨쥬게이트된다; 및
(d) 효소 기질을 첨가하여 고정된 샘플에 결합하는 항체의 양을 정량하는 단계.
CRP
발현값
측정에 적합한 프로토콜
(a) 샘플을 CRP에 특이적으로 결합하는 제1 항체와 인큐베이션하는 단계로, 상기 제 1항체는 고체상으로 고정된다;
(b) 세척하는 단계;
(c) CRP에 특이적으로 결합하는 제 2 항체를 첨가하는 단계로, 상기 제 2 항체는 효소와 컨쥬게이트된다; 및
(d) 효소 기질을 첨가하여 고정된 샘플에 결합하는 항체의 양을 정량하는 단계.
프로토콜 C:
CRP
발현값
측정에 적합한 프로토콜
(a)
지질 혼합물의 혼탁도를 측정하는 단계;
(b)
칼슘 존재하에서 샘플을 양을 아는 지질(바람직하게는 포스포릴콜린)과 접촉시키는 단계; 및
(c)
용액의 혼탁도를 측정하는 단계로, 상기 증가한 혼탁도는 CRP의 양과 관련된다.
실시예
9
TRAIL 및
IP
-10의 양을 분석하기 위한 ELISA의 상세한 설명
샘플 수집 및 저장 : 실온에 샘플의 노출은 최소화되어야 한다(6시간 미만). 혈청 분리 튜브(SST)를 사용하여 샘플을 원심분리(1200xg에서 5분간) 전에 적어도 30분동안 응고시킨다. 혈청을 즉시 분석하거나 분취하고 4-8 ℃에서 24시간까지 저장하거나 ≤ -20 ℃에서 3개월까지 보관할 수 있다. 반복된 동결-해동은 피해야 한다.
시약 준비 : 모든 시약은 사용 전에 실온으로 가져와야 한다.
기질 용액 : 컬러 시약 A와 B는 사용 후 10 분 이내에 같은 양으로 혼합해야 한다. 빛으로부터 보호해야 한다.
QC-1V, QC-2B 및 표준 : 모든 QC 및 표준을 해동하고 각 유리병(vial)에서 분리 표지된 폴리프로필렌 시험관으로 150uL을 옮겨야 한다. 사용 직후 -20 ℃로 다시 이동하여야 한다.
Trail 측정:
TRAIL 분석에 사용되는 물질을 하기 표 21에 나타내었다.
| 부분 | 설명 | 저장 상태 |
| TRAIL 마이크로플레이트 | 항 TRAIL 항체로 코팅된 96-웰 마이크로플레이트(8개 웰에 대해 12개 스트립) | 2-8 ℃ 에서 저장. |
|
TRAIL
접합체(Conjugate) |
방부제와 함께 서양 고추냉이 퍼옥시다아제와 컨쥬게이트된 항-TRAIL 특이적 항체 21ml | |
| Assay diluent MM1S | 방부제가 포함된 버퍼 단백질 베이스(buffered protein base) 11ml | |
| 세척 버퍼 농도 | 방부제를 포함한 버퍼된 계면활성제의 25배 농축액 21ml | |
| 컬러 시료 A | 안정화된 과산화수소 12 mL | |
| 컬러 시료 B | 테트라메틸벤지딘(tetramethylbenzidine, TMB) 12 mL | |
| 정지 용액 | 2 N 황산(sulfuric acid) 6 mL | |
| 플레이터 실러(Plate sealer) | 4 접착용 스트립(adhesive strips) | |
| 6 TRAIL 표준(Standards) | 500, 250, 125, 62.5, 31.2 및 0 [pg/mL] 농도에서 방부제를 포함하는 버퍼 단백질 베이스 내 재조합 인간 TRAIL 0.7ml를 함유하는 6개 유리병(vial) | 얻은 즉시 -20 ℃에서 저장 |
| QC -1V | 1ml | |
| QC -2B | 1ml |
TRAIL ELISA 절차
(a) 상기 언급된 샘플, 시약 및 표준을 준비한다.
(b) 플레이트 프레임으로부터 초과한 마이크로플레이트 스트립을 제거하고 건조팩이 함유된 호일 주머니(foil pouch )에 넣은 다음 다시 밀봉한다.
(c) 각 웰에 50 μL 의 Assay Diluent MM1S를 첨가한다.
(d) 웰당 50 μL의 표준, 샘플 또는 QC를 첨가한다. 제공된 접착용 스트립으로 씌운다.
(e) 550 rpm으로 설정된 마이크로플레이트 쉐어커(3 mm 궤도)에서 실온에서 30분동안 인큐베이트한다.
(f) 각 웰을 흡인하고 세척하는 것을 4번 반복한다. 세척 버퍼(300μL)를 각 웰에 가득 채워 세척한다. 세척 후, 남아있는 세척 버퍼를 흡인 건조하여 제거한다. 플레이트를 뒤집어 깨끗한 종이 타월로 닦아낸다.
(g) 각 웰에 200μL의 TRAIL 접합체를 첨가한다. 새 접착용 스트립으로 씌운다. 550 rpm으로 설정된 마이크로플레이트 쉐어커(3 mm 궤도)에서 실온에서 45분동안 인큐베이트한다.
(h) 단계에서 흡인/세척을 반복한다.
(i) 각 웰에 200μL의 기질 용액을 첨가한다. 실온에서 24-30분간 인큐베이트한다. 빛은 피한다.
(j) 각 웰에 50μL의 정지 용액을 첨가한다. 상기 웰의 컬러는 파란색에서 노란색으로 변해야 한다. 만약 웰에서 컬러가 녹색이거나 컬러 변화가 균일하지 않다면, 플레이트를 살짝 두드려 완전히 섞는다.
(k) 450nm로 설정된 마이크로플레이트 리더를 사용하여 각 웰의 광학 밀도를 즉시 결정한다. 파장을 보정하여 570nm로 설정하면 플레이트의 광학 불완전성을 보정할 수 있다.
TRAIL 농도 계산 : 각 샘플의 중복 판독값을 평균하고 평균 제로 표준 광학 밀도(O.D.)를 뺀다. 농도 (x 축)에 대한 각 표준 (y 축)의 평균 흡광도를 플로팅하여 표준 곡선을 만들고 최적 곡선을 그린다. 최소 r2는 0.96 이하가 되어서는 안된다. 낮은 r2 값이 나타나는 경우, 신뢰할 수 있는 결과를 얻으려면 실험을 반복해야 한다.
정밀도: 정밀도는 CLSI (이전의 NCCLS) EP05-A2 지침에 따라 평가되었다. 정밀도를 평가하기 위해 낮은 (11.4pg/ml), 중간 (58.8pg/ml) 및 높은 (539.0pg/ml) 생리학적 농도를 갖는 3 개의 샘플을 사용하였다. 결과는 표 22에 요약되어 있다. 여기서 Sr은 내부 실행 정밀도(within-run precision)이고 ST는 내부 장치 정밀도(within-device precision)이다:
| 낮은 (11.4 pg/ml) |
중간 (58.8pg/ml) |
높은 (539.0pg/ml) |
|
| # of runs | 18 | 18 | 18 |
| # of duplicates | 36 | 36 | 36 |
| Sr pg/mL | 0.84 | 2.45 | 13.2 |
| Sr CV (%) | 7.3% | 4.2% | 2.5% |
| ST pg/mL | 1.3 | 3.6 | 29.7 |
| ST CV (%) | 11.5% | 6.1% | 5.5% |
회복: TRAIL의 검출 가능한 레벨이 없는 5 개의 인간 혈청 샘플에 3 가지 레벨의 인간 재조합 TRAIL (250, 125 및 62.5pg/mL)을 첨가하여 회복을 평가하였다. 아래 표 23에 나와있는 것처럼 스파이크 값(spiked values)과 평균 회복값(average recovery)을 측정하고 계산하였다.
| 샘플 | 평균 회복 % | 범위 |
| 혈청 (n=5) | 77% | 75-78% |
선형성 : 분석법의 선형성을 평가하기 위해, 고농도 TRAIL을 함유한 5 개의 임상 샘플을 혈청 대체물을 사용하여 연속 희석하여 분석의 생리학적 범위 내의 값을 갖는 샘플을 생성하였다. 아래 표 24에 요약된 바와 같이 1:2, 1:4 및 1:8 희석에 대한 선형성은 각각 평균 97 %, 100 % 및 105 %였다.
| 혈청 (n=5) | ||
| 1:2 | 예상 평균 % | 97% |
| 범위 % | 90-104% | |
| 1:4 | 예상 평균 % | 100% |
| 범위 % | 90-108% | |
| 1:8 | 예상 평균 % | 105% |
| 범위 % | 90-121% |
민감도 : Blank of Limit (LOB)를 측정하기 위해 72 개의 혈청 대체 blank 샘플을 테스트하였다. Blank 샘플의 평균은 0.78 pg/ml이었고 표준 편차는 1.39 pg/ml이었다. 따라서 계산된 LOB는 3.07 pg/ml이다.
탐지 한계 (LOD)를 평가하기 위해 CLSI EP17-A 지침을 따랐다. 간단히 말해서, 미리 결정된 7 개의 농도에 대한 측정 분포가 특성화되었고, 각각 30 개의 독립적인 측정 (210 개의 측정)으로 10pg/ml의 LOD를 산출했다.
보정 :이 면역 분석법은 정제된 NS0 발현 재조합 인간 TRAIL에 대해 보정된다.
예상값 : 명백히 건강한 성인 (18세 초과)으로부터 얻은 샘플에서 TRAIL의 존재를 측정하였다. 범위 및 평균값은 표 25에 요약되어 있다.
| 샘플 유형 | 평균 pg/ml | 범위 pg/ml |
| 혈청 (n=34) | 90 | 17-157 |
교차 반응 및 간섭: 이 분석법은 자연적 TRAIL 및 재조합 인간 TRAIL을 인식한다. 4-1BB 리간드, APRIL, BAFF/BLyS, CD27 리간드, CD30 리간드, CD40 리간드, Fas 리간드, GITR 리간드, LIGHT, LT α1/β2, LT α2/β1, OPG, OX40 리간드, TNF-α, TNF-β, TRAIL R3, TRAIL R4, TRANCE 및 TWEAK를 혈청 대체에서 50 ng/mL로 제조하고 교차 반응성에 대해 분석하였다. 또한, 중간 범위의 재조합 인간 TRAIL 대조군에서 50 pg/mL의 이들 인자의 제조를 간섭에 대해 테스트하였다. 유의한 교차 반응이나 간섭은 관찰되지 않았다.
IP-10 측정: IP-10 분석에 사용된 물질은 아래 표 26에 나와 있다.
| 부분 | 설명 | 저장 상태 |
| IP -10 마이크로플레이트 | 항 IP-10 항체로 코팅된 96-웰 마이크로플레이트(8개 웰에 대해 12개 스트립) | 2-8 ℃ 에서 저장. |
|
IP
-10
접합체(Conjugate) |
방부제와 함께 서양 고추냉이 퍼옥시다아제와 컨쥬게이트된 항-IP-10 특이적 항체 21ml | |
| Assay diluent MM56 | 방부제가 포함된 버퍼 단백질 베이스(buffered protein base) 11ml | |
| 세척 버퍼 농도 | 방부제를 포함한 버퍼된 계면활성제의 25배 농축액 21ml | |
| 컬러 시료 A | 안정화된 과산화수소 12 mL | |
| 컬러 시료 B | 테트라메틸벤지딘(tetramethylbenzidine, TMB) 12 mL | |
| 정지 용액 | 2 N 황산(sulfuric acid) 6 mL | |
| 플레이터 실러(Plate sealer) | 4 접착용 스트립(adhesive strips) | |
| 6 IP -10 표준(Standards) | 1000, 500, 250, 125, 62.5 및 0 [pg/mL] 농도에서 방부제를 포함하는 버퍼 단백질 베이스 내 재조합 인간 IP-10 0.7ml를 함유하는 6개 유리병(vial) | 얻은 즉시 -20 ℃에서 저장 |
| QC -1V | 1ml | |
| QC -2B | 1ml |
IP
-10 ELISA 절차
a) 상기 언급된 샘플, 시약 및 표준을 준비한다.
b) 플레이트 프레임으로부터 초과한 마이크로플레이트 스트립을 제거하고 건조팩이 함유된 호일 주머니(foil pouch )에 넣은 다음 다시 밀봉한다.
c) 각 웰에 50 μL 의 Assay Diluent MM56를 첨가한다.
d) 웰당 50 μL의 표준, 샘플 또는 QC를 첨가한다. 제공된 접착용 스트립으로 씌운다.
e) 550 rpm으로 설정된 마이크로플레이트 쉐어커(3 mm 궤도)에서 실온에서 30분동안 인큐베이트한다.
f) 각 웰을 흡인하고 세척하는 것을 4번 반복한다. 세척 버퍼(300μL)를 각 웰에 가득 채워 세척한다. 세척 후, 남아있는 세척 버퍼를 흡인 건조하여 제거한다. 플레이트를 뒤집어 깨끗한 종이 타월로 닦아낸다.
g) 각 웰에 200μL의 TRAIL 접합체를 첨가한다. 새 접착용 스트립으로 씌운다. 550 rpm으로 설정된 마이크로플레이트 쉐어커(3 mm 궤도)에서 실온에서 45분동안 인큐베이트한다.
h) (g) 단계에서 흡인/세척을 반복한다.
i) 각 웰에 200μL의 기질 용액을 첨가한다. 실온에서 10분간 인큐베이트한다. 빛은 피한다.
j) 각 웰에 50μL의 정지 용액을 첨가한다. 상기 웰의 컬러는 파란색에서 노란색으로 변해야 한다. 만약 웰에서 컬러가 녹색이거나 컬러 변화가 균일하지 않다면, 플레이트를 살짝 두드려 완전히 섞는다.
k) 450nm로 설정된 마이크로플레이트 리더를 사용하여 각 웰의 광학 밀도를 즉시 결정한다. 파장을 보정하여 570nm로 설정하면 플레이트의 광학 불완전성을 보정할 수 있다.
IP -10 농도 계산 : 각 샘플의 중복 판독값을 평균하고 평균 제로 표준 광학 밀도(O.D.)를 뺀다. 농도 (x 축)에 대한 각 표준 (y 축)의 평균 흡광도를 플로팅하여 표준 곡선을 만들고 최적 곡선을 그린다. 최소 r2는 0.96 이하가 되어서는 안된다. 낮은 r2 값이 나타나는 경우, 신뢰할 수 있는 결과를 얻으려면 실험을 반복해야 한다.
정밀도: 정밀도는 CLSI (이전의 NCCLS) EP05-A2 지침에 따라 평가되었다. 정밀도를 평가하기 위해 낮은 (69.4pg/ml), 중간 (228.2pg/ml) 및 높은 (641.5pg/ml) 생리학적 농도를 갖는 3 개의 샘플을 사용하였다. 결과는 표 27에 요약되어 있다. 여기서 Sr은 내부 실행 정밀도(within-run precision)이고 ST는 내부 장치 정밀도(within-device precision)이다:
| 낮은 (69.4 pg/ml) |
중간 (228.2 pg/ml) |
높은 (641.5 pg/ml) |
|
| # of runs | 18 | 18 | 18 |
| # of duplicates | 36 | 36 | 36 |
| Sr pg/mL | 4.0 | 5.6 | 21.1 |
| Sr CV (%) | 5.8% | 2.4% | 3.3% |
| ST pg/mL | 4.9 | 12.9 | 37.2 |
| ST CV (%) | 7.1% | 5.7% | 5.8% |
회복: IP-10의 검출 가능한 레벨이 없는 5 개의 인간 혈청 샘플에 3 가지 레벨의 인간 IP-10 (500, 250 및 125pg/mL)을 첨가하여 회복을 평가하였다. 아래 표 28에 나와있는 것처럼 스파이크 값(spiked values)과 평균 회복값(average recovery)을 측정하고 계산하였다.
| 샘플 | 평균 회복 % | 범위 |
| 혈청/혈장 (n=5) | 77 | 72-80% |
선형성 : IP-10 분석의 선형성을 평가하기 위해, 873.7 내지 1110.4pg/mL범위의 고농도 IP-10을 함유한 5 개의 임상 샘플을 혈청 대체물을 사용하여 연속 희석하여 분석의 생리학적 범위 내의 값을 갖는 샘플을 제조하였다. 아래 표 29에 요약된 바와 같이 1:2, 1:4 및 1:8 희석에 대한 선형성은 각각 평균 98 %, 102 % 및 104 %였다.
| 혈청 (n=5) | ||
| 1:2 | 예상 평균 % | 98% |
| 범위 % | 93-102% | |
| 1:4 | 예상 평균 % | 102% |
| 범위 % | 97-107% | |
| 1:8 | 예상 평균 % | 104% |
| 범위 % | 96-111% |
민감도 : Blank of Limit (LOB)를 측정하기 위해 72 개의 혈청 대체 blank 샘플을 테스트하였다. Blank 샘플의 평균은 0.23 pg/ml이었고 표준 편차는 1.26 pg/ml이었다. 따라서 계산된 LOB는 2.29 pg/ml이다.
탐지 한계 (LOD)를 평가하기 위해 CLSI EP17-A 지침을 따랐다. 간단히 말해서, 미리 결정된 7 개의 농도에 대한 측정 분포가 특성화되었고, 각각 30 개의 독립적인 측정 (210 개의 측정)으로 10pg/ml의 LOD를 산출했다.
보정(Calibration) :이 면역 분석법은 고도 정제된 대장균 발현 재조합 인간 IP-10에 대해 보정된다.
예상값 : 명백히 건강한 성인으로부터 얻은 샘플에서 IP-10의 존재를 측정하였다. 범위 및 평균값은 표 30에 요약되어 있다.
| 샘플 유형 | 평균 pg/ml | 범위 pg/ml |
| 혈청 (n=34) | 119 | 29-525 |
교차 반응 및 간섭: 이 분석법은 자연적 IP-10 및 재조합 인간 TRAIL을 인식한다. BLC/BCA-1, ENA-78, GCP-2, GROα, GRO γ, IFN-γ, IL-8, I-TAC, MIG, NAP-2, SDF-1α 및 SDF-1β를 혈청 대체에서 50 ng/mL로 제조하고 교차 반응성에 대해 분석하였다. 또한, 중간 범위의 재조합 인간 IP-10 대조군에서 50 pg/mL의 이들 인자의 제조를 간섭에 대해 테스트하였다. 유의한 교차 반응이나 간섭은 관찰되지 않았다.
실시예
10
TRAIL 및 질병 진단
환자의 예후, 질병의 중증도 및 결과를 평가하는 것이 종종 임상적으로 유용하다. 본 발명자들은 낮은 레벨의 TRAIL은 환자의 예후 및 결과가 나쁘고 질병의 심각도가 높음과 유의한 상관 관계가 있음을 발견했다. 예를 들어, 중환자실 (ICU)의 성인 환자는 감염 또는 비 감염 원인에 관계없이 병세가 적은 다른 모든 환자와 비교하여 TRAIL 레벨이 유의하게 낮았다. 중간 혈청 농도는 각각 9pg/ml 대 80pg (ranksum P <0.001, 도 36a)이었다.
TRAIL 혈청 농도를 3 개월에서 5 세 사이의 40 명의 네덜란드 소아 환자에서 측정하였다. 결국 ICU로 결정된 (질병 합병증 및 예후가 좋지 않음)환자 또는 심지어 사망한 환자는 나머지 환자들과 비교하여 TRAIL 혈청 농도가 유의하게 낮았다 (중간값 11 대 85, ranksum P <0.001). 놀랍게도 가장 낮은 TRAIL 레벨 (<5pg/ml)은 전체 집단에서 사망한 유일한 두 자녀에게서 측정되었다. 이러한 결과는 TRAIL이 질병의 중증도와 결과를 예측하는 예후 지표로서 사용될 수 있음을 나타낸다.
실시예
11
TRAIL 연령 및 성별 파라미터
건강한 개인 또는 비 감염성 질병 환자에서의 TRAIL의 기본 레벨은 임신 가능 연령의 남성과 비교하여 여성에서 더 낮지만(t-test P<0.001) (도 37a), 임신 가능 연령 전 또는 후 연령에서 불변하였다(t-test P=0.9, 도 37b). 이러한 경향은 감염성 질병이 있는 환자에서는 관찰되지 않았다.
실시예
12
예시적인 매니폴드(Manifolds), 초평면(Hyperplanes) 및 좌표(Coordinates)
1 차원
매니폴드(One-dimensional Manifold)
n=1 인 경우 매니폴드 S는 곡선이고 초평면 π는 단일 방향 δ 1을 정의하는 한 축이다. 본 실시예의 좌표 δ1은 선택적으로 및 바람직하게는 폴리펩타이드 D1, D2 등의 선형 조합 b 0+b 1D1+b 2D2+...,이다.
아래의 표 31은 n=1에 대해 획득된 진단 성능 (AUC)을 나열한다. 성능은 각 행에 지정된 집단에 대해 leave-10 % 교차 검증을 사용하여 계산되었다. 1-4 행에서 분석 대상체는 박테리아성 또는 바이러스성 감염이 있었고 확률 분류 함수 f(δ1)가 대상체의 박테리아성 감염 가능성을 나타낼 수 있도록 좌표 δ1을 계산하였다. 5-8 행에서 분석 대상체는 감염성 또는 비 감염성이었고 확률 분류 함수 f(δ1)가 대상체의 감염되었을 가능성을 나타낼 수 있도록 좌표 δ1을 계산하였다. 10-12 행에서 분석 대상체는 박테리아성 또는 비-박테리아성 감염이 있었고 확률 분류 함수 f(δ1)가 대상체의 박테리아성 감염 가능성을 나타낼 수 있도록 좌표 δ1을 계산하였다. 1-4 행에서 P 및 N 열은 각각 박테리아성 및 바이러스성 환자의 수에 해당하며, 5-8 행에서 P 및 N 열은 각각 감염성 및 비-감염성 환자의 수에 해당한다. 9-12 행에서, P 및 N 열은 박테리아성 및 비-박테리아성 환자의 수에 각각 대응한다. 대다수 및 만장일치 집단은 검증된 모델에 대한 집단 유형을 나타낸다.
| No. | 집단 | 폴리펩타이드 | AUC | P | N | ||
| 1 | 대다수 | CRP | TRAIL | 0.93 | 319 | 334 | |
| 2 | 대다수 | CRP | IP-10 | TRAIL | 0.94 | 319 | 334 |
| 3 | 만장일치 | CRP | TRAIL | 0.95 | 256 | 271 | |
| 4 | 만장일치 | CRP | IP-10 | TRAIL | 0.96 | 256 | 271 |
| 5 | 대다수 | CRP | TRAIL | 0.93 | 653 | 112 | |
| 6 | 대다수 | CRP | IP-10 | TRAIL | 0.96 | 653 | 112 |
| 7 | 만장일치 | CRP | TRAIL | 0.93 | 527 | 112 | |
| 8 | 만장일치 | CRP | IP-10 | TRAIL | 0.97 | 527 | 112 |
| 9 | 대다수 | CRP | TRAIL | 0.94 | 319 | 446 | |
| 10 | 대다수 | CRP | IP-10 | TRAIL | 0.94 | 319 | 446 |
| 11 | 만장일치 | CRP | TRAIL | 0.95 | 256 | 383 | |
| 12 | 만장일치 | CRP | IP-10 | TRAIL | 0.96 | 256 | 383 |
아래 표 32는 표 31에 열거된 12 가지 경우 각각에 대해 좌표 δ1을 정의하는데 사용된 계수 b 0, b 1, b 2 등을 열거한다. 왼쪽의 첫 번째 계수는 b 0이고, 왼쪽에서 오른쪽으로 계수는 표 28의 각 행에 있는 폴리펩타이드의 순서에 해당한다. 계수는 다음의 각 폴리펩타이드에 대한 농도 눈금에 해당한다 : TRAIL (pg/ml) , IP-10 (pg/ml) 및 CRP (μg/ml).
주어진 폴리펩타이드 세트에 대해, 얻어진 계수는 상이한 집단 사이에서 작은 편차를 갖는다. 그럼에도 불구하고, 본 실시예의 확률 분류 함수 및 좌표에 대한 계수는 대다수 집단에 대해 획득된 것과 상응하는 것이 바람직하다.
| No. | 계수 | |||
| 1 | 0.64814 | 0.027472 | -0.029953 | |
| 2 | 0.71542 | 0.028119 | -0.00028168 | -0.029013 |
| 3 | 0.636 | 0.034565 | -0.033669 | |
| 4 | 0.79543 | 0.035748 | -0.00058691 | -0.03195 |
| 5 | -2.0549 | 0.17237 | 0.016837 | |
| 6 | -2.3344 | 0.1263 | 0.00592 | 0.005213 |
| 7 | -2.3469 | 0.16625 | 0.018624 | |
| 8 | -2.7949 | 0.12261 | 0.0061124 | 0.0079169 |
| 9 | -0.08503 | 0.034954 | -0.027839 | |
| 10 | -0.088207 | 0.034878 | 2.2524e-05 | -0.027916 |
| 11 | -0.26606 | 0.044289 | -0.030997 | |
| 12 | -0.23907 | 0.044938 | -0.00018635 | -0.03042 |
아래 표 33은 1 차원 매니폴드에 대해 달성된 진단 성능 (AUC)을 열거한다. 성능은 대다수 집단에 대한 leave-10%-out cross validation을 사용하여 계산되었다. 1-55 행에서, 분석 대상체는 박테리아성 또는 바이러스성 감염이 있었고 확률 분류 함수 f(δ1)는 테스트 대상체의 박테리아성 감염 가능성을 나타냈다. 56-110 행에서 분석 대상체는 감염성 또는 비-감염성이었고 확률 분류 함수 f(δ1)는 대상체의 감염 가능성을 나타 냈다. 1-55 행에서 P 및 N 열은 각각 박테리아성 및 바이러스성 환자의 수에 해당하고 56-110 행에서 P 및 N 열은 각각 감염성 환자 및 비-감염 환자의 수에 해당한다.
| No. | 폴리펩타이드 | AUC | P | N | |||||
| 1 | CRP | IL1ra | 0.88 | 142 | 141 | ||||
| 2 | CRP | IP-10 | 0.90 | 295 | 299 | ||||
| 3 | CRP | PCT | 0.87 | 51 | 50 | ||||
| 4 | CRP | SAA | 0.90 | 255 | 241 | ||||
| 5 | IL1ra | IP-10 | 0.64 | 142 | 142 | ||||
| 6 | IL1ra | PCT | 0.62 | 19 | 14 | ||||
| 7 | IL1ra | SAA | 0.83 | 124 | 122 | ||||
| 8 | IL1ra | TRAIL | 0.88 | 142 | 142 | ||||
| 9 | IP-10 | PCT | 0.74 | 51 | 49 | ||||
| 10 | IP-10 | SAA | 0.85 | 251 | 242 | ||||
| 11 | IP-10 | TRAIL | 0.88 | 295 | 297 | ||||
| 12 | PCT | SAA | 0.78 | 45 | 40 | ||||
| 13 | PCT | TRAIL | 0.87 | 51 | 50 | ||||
| 14 | SAA | TRAIL | 0.90 | 255 | 244 | ||||
| 15 | CRP | IL1ra | IP-10 | 0.90 | 142 | 141 | |||
| 16 | CRP | IL1ra | PCT | 0.82 | 19 | 14 | |||
| 17 | CRP | IL1ra | SAA | 0.89 | 124 | 121 | |||
| 18 | CRP | IL1ra | TRAIL | 0.94 | 142 | 141 | |||
| 19 | CRP | IP-10 | PCT | 0.89 | 51 | 49 | |||
| 20 | CRP | IP-10 | SAA | 0.91 | 251 | 239 | |||
| 21 | CRP | PCT | SAA | 0.88 | 45 | 40 | |||
| 22 | CRP | PCT | TRAIL | 0.93 | 51 | 50 | |||
| 23 | CRP | SAA | TRAIL | 0.94 | 255 | 241 | |||
| 24 | IL1ra | IP-10 | PCT | 0.62 | 19 | 14 | |||
| 25 | IL1ra | IP-10 | SAA | 0.85 | 124 | 122 | |||
| 26 | IL1ra | IP-10 | TRAIL | 0.88 | 142 | 142 | |||
| 27 | IL1ra | PCT | SAA | 0.76 | 17 | 13 | |||
| 28 | IL1ra | PCT | TRAIL | 0.71 | 19 | 14 | |||
| 29 | IL1ra | SAA | TRAIL | 0.92 | 124 | 122 | |||
| 30 | IP-10 | PCT | SAA | 0.81 | 45 | 39 | |||
| 31 | IP-10 | PCT | TRAIL | 0.86 | 51 | 49 | |||
| 32 | IP-10 | SAA | TRAIL | 0.91 | 251 | 242 | |||
| 33 | PCT | SAA | TRAIL | 0.86 | 45 | 40 | |||
| 34 | CRP | IL1ra | IP-10 | PCT | 0.83 | 19 | 14 | ||
| 35 | CRP | IL1ra | IP-10 | SAA | 0.92 | 124 | 121 | ||
| 36 | CRP | IL1ra | IP-10 | TRAIL | 0.94 | 142 | 141 | ||
| 37 | CRP | IL1ra | PCT | SAA | 0.74 | 17 | 13 | ||
| 38 | CRP | IL1ra | PCT | TRAIL | 0.90 | 19 | 14 | ||
| 39 | CRP | IL1ra | SAA | TRAIL | 0.94 | 124 | 121 | ||
| 40 | CRP | IP-10 | PCT | SAA | 0.88 | 45 | 39 | ||
| 41 | CRP | IP-10 | PCT | TRAIL | 0.92 | 51 | 49 | ||
| 42 | CRP | IP-10 | SAA | TRAIL | 0.94 | 251 | 239 | ||
| 43 | CRP | PCT | SAA | TRAIL | 0.92 | 45 | 40 | ||
| 44 | IL1ra | IP-10 | PCT | SAA | 0.70 | 17 | 13 | ||
| 45 | IL1ra | IP-10 | PCT | TRAIL | 0.70 | 19 | 14 | ||
| 46 | IL1ra | IP-10 | SAA | TRAIL | 0.91 | 124 | 122 | ||
| 47 | IL1ra | PCT | SAA | TRAIL | 0.82 | 17 | 13 | ||
| 48 | IP-10 | PCT | SAA | TRAIL | 0.85 | 45 | 39 | ||
| 49 | CRP | IL1ra | IP-10 | PCT | SAA | 0.82 | 17 | 13 | |
| 50 | CRP | IL1ra | IP-10 | PCT | TRAIL | 0.75 | 19 | 14 | |
| 51 | CRP | IL1ra | IP-10 | SAA | TRAIL | 0.94 | 124 | 121 | |
| 52 | CRP | IL1ra | PCT | SAA | TRAIL | 0.78 | 17 | 13 | |
| 53 | CRP | IP-10 | PCT | SAA | TRAIL | 0.92 | 45 | 39 | |
| 54 | IL1ra | IP-10 | PCT | SAA | TRAIL | 0.62 | 17 | 13 | |
| 55 | CRP | IL1ra | IP-10 | PCT | SAA | TRAIL | 0.74 | 17 | 13 |
| 56 | CRP | IL1ra | 0.91 | 283 | 87 | ||||
| 57 | CRP | IP-10 | 0.96 | 594 | 102 | ||||
| 58 | CRP | PCT | 0.85 | 101 | 6 | ||||
| 59 | CRP | SAA | 0.91 | 496 | 78 | ||||
| 60 | IL1ra | IP-10 | 0.89 | 284 | 87 | ||||
| 61 | IL1ra | PCT | 0.79 | 33 | 6 | ||||
| 62 | IL1ra | SAA | 0.91 | 246 | 64 | ||||
| 63 | IL1ra | TRAIL | 0.86 | 284 | 87 | ||||
| 64 | IP-10 | PCT | 0.73 | 100 | 6 | ||||
| 65 | IP-10 | SAA | 0.96 | 493 | 81 | ||||
| 66 | IP-10 | TRAIL | 0.91 | 592 | 107 | ||||
| 67 | PCT | SAA | 0.89 | 85 | 3 | ||||
| 68 | PCT | TRAIL | 0.60 | 101 | 7 | ||||
| 69 | SAA | TRAIL | 0.93 | 499 | 81 | ||||
| 70 | CRP | IL1ra | IP-10 | 0.95 | 283 | 87 | |||
| 71 | CRP | IL1ra | PCT | 0.76 | 33 | 6 | |||
| 72 | CRP | IL1ra | SAA | 0.92 | 245 | 64 | |||
| 73 | CRP | IL1ra | TRAIL | 0.93 | 283 | 87 | |||
| 74 | CRP | IP-10 | PCT | 0.81 | 100 | 6 | |||
| 75 | CRP | IP-10 | SAA | 0.97 | 490 | 78 | |||
| 76 | CRP | PCT | SAA | 0.88 | 85 | 3 | |||
| 77 | CRP | PCT | TRAIL | 0.87 | 101 | 6 | |||
| 78 | CRP | SAA | TRAIL | 0.95 | 496 | 78 | |||
| 79 | IL1ra | IP-10 | PCT | 0.77 | 33 | 6 | |||
| 80 | IL1ra | IP-10 | SAA | 0.94 | 246 | 64 | |||
| 81 | IL1ra | IP-10 | TRAIL | 0.90 | 284 | 87 | |||
| 82 | IL1ra | PCT | SAA | 0.72 | 30 | 3 | |||
| 83 | IL1ra | PCT | TRAIL | 0.67 | 33 | 6 | |||
| 84 | IL1ra | SAA | TRAIL | 0.90 | 246 | 64 | |||
| 85 | IP-10 | PCT | SAA | 0.98 | 84 | 3 | |||
| 86 | IP-10 | PCT | TRAIL | 0.68 | 100 | 6 | |||
| 87 | IP-10 | SAA | TRAIL | 0.96 | 493 | 81 | |||
| 88 | PCT | SAA | TRAIL | 0.98 | 85 | 3 | |||
| 89 | CRP | IL1ra | IP-10 | PCT | 0.77 | 33 | 6 | ||
| 90 | CRP | IL1ra | IP-10 | SAA | 0.95 | 245 | 64 | ||
| 91 | CRP | IL1ra | IP-10 | TRAIL | 0.95 | 283 | 87 | ||
| 92 | CRP | IL1ra | PCT | SAA | 0.73 | 30 | 3 | ||
| 93 | CRP | IL1ra | PCT | TRAIL | 0.74 | 33 | 6 | ||
| 94 | CRP | IL1ra | SAA | TRAIL | 0.92 | 245 | 64 | ||
| 95 | CRP | IP-10 | PCT | SAA | 0.98 | 84 | 3 | ||
| 96 | CRP | IP-10 | PCT | TRAIL | 0.77 | 100 | 6 | ||
| 97 | CRP | IP-10 | SAA | TRAIL | 0.97 | 490 | 78 | ||
| 98 | CRP | PCT | SAA | TRAIL | 0.80 | 85 | 3 | ||
| 99 | IL1ra | IP-10 | PCT | SAA | 0.91 | 30 | 3 | ||
| 100 | IL1ra | IP-10 | PCT | TRAIL | 0.67 | 33 | 6 | ||
| 101 | IL1ra | IP-10 | SAA | TRAIL | 0.94 | 246 | 64 | ||
| 102 | IL1ra | PCT | SAA | TRAIL | 0.78 | 30 | 3 | ||
| 103 | IP-10 | PCT | SAA | TRAIL | 0.65 | 84 | 3 | ||
| 104 | CRP | IL1ra | IP-10 | PCT | SAA | 0.91 | 30 | 3 | |
| 105 | CRP | IL1ra | IP-10 | PCT | TRAIL | 0.66 | 33 | 6 | |
| 106 | CRP | IL1ra | IP-10 | SAA | TRAIL | 0.95 | 245 | 64 | |
| 107 | CRP | IL1ra | PCT | SAA | TRAIL | 0.73 | 30 | 3 | |
| 108 | CRP | IP-10 | PCT | SAA | TRAIL | 0.97 | 84 | 3 | |
| 109 | IL1ra | IP-10 | PCT | SAA | TRAIL | 0.78 | 30 | 3 | |
| 110 | CRP | IL1ra | IP-10 | PCT | SAA | TRAIL | 0.73 | 30 | 3 |
아래 표 34은 표 33에 열거된 110 가지 경우 각각에 대해 좌표 δ1를 정의하는데 사용된 계수 b 0, b 1, b 2 등을 열거한다. 왼쪽의 첫 번째 계수는 b 0이고, 왼쪽에서 오른쪽으로 계수는 표 33의 각 행에 있는 폴리펩타이드의 순서에 해당한다. 계수는 다음의 각 폴리펩타이드에 대한 농도 눈금에 해당한다: TRAIL (pg/ml) , IP-10 (pg/ml), CRP (μg/ml), PCT (ng/ml), SAA (g/ml) 및 IL1ra (g/ml)
| No. | 계수 | ||||||
| 1 | -1.997 | 0.0363 | -9849178.8 | ||||
| 2 | -1.6069 | 0.039722 | -0.0009 | ||||
| 3 | -2.9681 | 0.054137 | 0.6405 | ||||
| 4 | -2.33196 | 0.034353 | 1098.3777 | ||||
| 5 | 0.4715979 | 47954608.09 | -0.00089 | ||||
| 6 | -0.74822 | -69280395.624 | 4.5607 | ||||
| 7 | -1.706206 | -33345728.8342 | 5283.68 | ||||
| 8 | 3.0601663 | 43833567.7377 | -0.03151 | ||||
| 9 | -0.13268 | -0.00060898 | 0.86013 | ||||
| 10 | -1.01872 | -0.0009684361 | 4677.8311 | ||||
| 11 | 2.5632 | 0.00031349 | -0.0288 | ||||
| 12 | -1.35195 | 1.1895403 | 2349.8702 | ||||
| 13 | 1.4742 | 0.4382 | -0.019169 | ||||
| 14 | 1.08972 | 2962.7685 | -0.02176 | ||||
| 15 | -1.27532 | 0.039986 | 6.264E+7 | -0.00165 | |||
| 16 | -2.3376 | 0.0475326 | -8.42E+7 | 1.07655 | |||
| 17 | -2.23709 | 0.027867 | -2.22E+7 | 2098.4 | |||
| 18 | 0.9001561 | 0.030146 | 2.0497E+7 | -0.0266 | |||
| 19 | -2.5383 | 0.051698 | -0.0005 | 0.65349 | |||
| 20 | -1.6940577 | 0.034481544 | -0.00109 | 1378.2 | |||
| 21 | -2.7487888 | 0.054245413 | 1.4735726 | -1243.01 | |||
| 22 | -1.421 | 0.04535 | 0.42793 | -0.010529 | |||
| 23 | 0.1044034 | 0.0312776 | 183.3117 | -0.01891 | |||
| 24 | -0.0013248 | -4107077 | -0.001241 | 4.8755 | |||
| 25 | -1.054077 | 21179055 | -0.001377 | 5777 | |||
| 26 | 3.0605 | 43882108 | -1.118-06 | -0.03151 | |||
| 27 | -1.9806377 | -68741718 | 2.91 | 4823 | |||
| 28 | 2.844483 | 113905139.6 | 1.941 | -0.0342 | |||
| 29 | 1.4399 | -7296968.1 | 3745.49 | -0.0264 | |||
| 30 | -0.8562752 | -0.000765 | 1.3263344 | 2427.6 | |||
| 31 | 1.357 | 0.00045394 | 0.38993 | -0.020588 | |||
| 32 | 1.0917705 | -0.000163 | 3048.4182 | -0.021174 | |||
| 33 | 0.48274 | 0.89320046 | 1431.011 | -0.013629 | |||
| 34 | -0.6620 | 0.07214 | 75533424 | -0.003888 | 1.5 | ||
| 35 | -1.433018 | 0.034006 | 59894763 | -0.002 | 2425.771 | ||
| 36 | 1.074937 | 0.03259 | 50294164 | -0.00084 | -0.0251 | ||
| 37 | -2.302 | 0.038 | -70994467 | 1.1316 | 893.395 | ||
| 38 | 1.903272 | 0.061632 | -81575254 | -0.084 | -0.0477 | ||
| 39 | 0.65146 | 0.025 | 10145313 | 1236 | -0.02483 | ||
| 40 | -2.32016 | 0.04984696 | -5.5688E-4 | 1.528887 | -949.2 | ||
| 41 | -1.4662 | 0.045264 | 0.00021523 | 0.40033 | -0.011113 | ||
| 42 | 0.14333 | 0.03169 | -0.0003975 | 329.7448 | -0.0177 | ||
| 43 | -1.109923 | 0.050385 | 1.24 | -1930.2 | -0.011 | ||
| 44 | -1.087150 | 2715886 | -0.002014 | 4.286 | 6082.17 | ||
| 45 | 2.9154 | -1508120 | 0.00092636 | 2.126 | -0.0397 | ||
| 46 | 1.55114 | 17100158 | -0.00062 | 4082.939 | -0.0252 | ||
| 47 | 2.85226 | -27909258 | 0.68134 | 7639.7 | -0.0560 | ||
| 48 | 0.446 | 6.13e-05 | 0.87764371 | 1423.99 | -0.0134 | ||
| 49 | -0.3021 | 0.07676 | 172681901 | -0.00652 | 1.250 | 4736.86 | |
| 50 | 1.937 | 0.0671 | -4.62E+7 | -0.000873 | -0.121 | -0.044 | |
| 51 | 0.78878 | 0.029267 | 54069432 | -0.00134 | 1576.6 | -0.0219 | |
| 52 | 2.309 | 0.041577 | -74159142 | -0.098620 | 3598 | -0.055 | |
| 53 | -1.1542 | 0.0512 | 0.00023 | 1.188 | -2055 | -0.0116 | |
| 54 | 3.2 | 14035678 | -0.0012627 | 1.03 | 8903.82 | -0.055 | |
| 55 | 5.59 | 0.10 | 1.062E+8 | -0.009695 | -0.687 | 14133 | -0.078 |
| 56 | -1.021759 | 0.11089 | 3.996E+8 | ||||
| 57 | -1.9467 | 0.11347 | 0.0063336 | ||||
| 58 | -84.98948 | 0.0639025 | 860.3249 | ||||
| 59 | -0.3299621 | 0.091563631 | 9898.8177 | ||||
| 60 | -1.0006445 | 107920251.6624 | 0.00721 | ||||
| 61 | -41.585735 | 596535240 | 419.2 | ||||
| 62 | -0.4789050 | 234257296.8937 | 14320 | ||||
| 63 | 0.09918792 | 812307573.5455 | 0.00066 | ||||
| 64 | -107.18015 | 0.00069423293 | 1089.4251 | ||||
| 65 | -2.05501 | 0.00967490979 | 12590.5 | ||||
| 66 | 0.19189 | 0.0092076 | -0.00905 | ||||
| 67 | -11.30895 | 122.7205081 | 165893.71 | ||||
| 68 | 0.98581 | 6.5788 | 0.0041105 | ||||
| 69 | -1.366750 | 19453.2163 | 0.010541 | ||||
| 70 | -2.301980 | 0.10876 | -77782071 | 0.0062 | |||
| 71 | -39.915 | 0.048935 | 559628637 | 393.7 | |||
| 72 | -0.885780 | 0.0663 | 244256710 | 8656.83 | |||
| 73 | -2.694252 | 0.142003 | 157875482 | 0.0129 | |||
| 74 | -84.66684 | 0.07831107 | 0.0014 | 846.608 | |||
| 75 | -2.5885198 | 0.081369191 | 0.00927 | 5900.1661 | |||
| 76 | -10.12169 | 0.06793071342 | 108.84 | 131629 | |||
| 77 | -82.88872 | 0.08303337 | 822.6365 | 0.011421 | |||
| 78 | -2.33978 | 0.106214424 | 10662.5415 | 0.013257 | |||
| 79 | -41.369532 | 744190123.3893 | -0.000381 | 417.43 | |||
| 80 | -2.266204 | -130390666 | 0.0091619 | 12128 | |||
| 81 | -0.50010 | 82287681 | 0.007583 | -0.005459 | |||
| 82 | -2.47028 | 6837963488 | -8.1908 | 377360 | |||
| 83 | -41.5502 | 560182293 | 418.212 | 0.00099 | |||
| 84 | -1.8577 | 29398797 | 17111.2 | 0.011194 | |||
| 85 | -156.16 | 0.4328 | 28.96307 | 21649017 | |||
| 86 | -107.01823 | 0.00029753173 | 1086.1672 | 0.00330 | |||
| 87 | -2.0464 | 0.00969 | 12572.6828 | -9.3941e-05 | |||
| 88 | -2614.99 | 471.6 | 80696477 | 24.2 | |||
| 89 | -39.82 | 0.0491 | 611767730 | -0.0001 | 392.929 | ||
| 90 | -2.81351 | 0.070555 | -157521601 | 0.00937 | 6854 | ||
| 91 | -2.9416 | 0.118 | -61236289 | 0.00552 | 0.005871 | ||
| 92 | -2.349 | -0.07356 | 7107285383 | -8.6576 | 403954 | ||
| 93 | -38.781 | 0.05763 | 383823513 | 373.75 | 0.00857 | ||
| 94 | -2.782 | 0.0965657 | -4665192.1 | 9692.125 | 0.013998 | ||
| 95 | -132.7427 | 10.567847038 | 0.3202 | -132.70 | 4998296 | ||
| 96 | -83.41464 | 0.08349426 | 0.000498 | 827.6066 | 0.00927 | ||
| 97 | -2.9639303 | 0.084631596 | 0.008696 | 6461.9905 | 0.00369 | ||
| 98 | 1.185E+14 | 9037614498892 | -1.05E+14 | 4.83248e+18 | 2.32E+12 | ||
| 99 | -99.64 | 116933544267 | 0.196688 | -296 | 9471186 | ||
| 100 | -41.177966 | 713679677.7954 | -0.00058 | 413.88 | 0.002761 | ||
| 101 | -2.6391378 | -124943185 | 0.0088176 | 12684.8 | 0.00349 | ||
| 102 | -163.16785 | 68076716508 | -272.0191 | 8853215 | 1.3718 | ||
| 103 | -134.8402 | 0.09197 | 24.21772 | 11007611 | 0.9352 | ||
| 104 | -82.7 | 5.367 | 32157214873 | 0.1975318 | -195 | 5448434 | |
| 105 | -35.325 | 0.0803 | 823767988 | -0.002344 | 327.2 | 0.024158 | |
| 106 | -3.579 | 0.080040 | -151905670 | 0.008791 | 7390.9 | 0.0065 | |
| 107 | -338.972 | 29.2 | 43593035460 | -106.418 | -1129873 | 2.78 | |
| 108 | -237.8248 | 8.2370 | 0.4843 | -22.217 | -96788.08 | 1.563 | |
| 109 | -8.51E+14 | -5.96133e+22 | 3.97E+12 | 2.798E+13 | 1.757e+18 | 4.06E+12 | |
| 110 | -276.3 | 9.549 | -4575512593 | 0.58062 | -16.687 | -9.83E+5 | 1.839 |
2차원 매니폴드(Two-dimensional Manifold)
n=2 인 경우 매니폴드 S는 곡면이고 초평면 π는 제 1 방향 δ 0 및 제 2 방향 δ 1으로 정의되는 한 평면이다. 본 실시예의 좌표 δ0은 선택적으로 및 바람직하게는 폴리펩타이드 D1, D2 등의 선형 조합 a0+a 1D1+a 2D2+...,이고, 좌표 δ1은 선택적으로 및 바람직하게는 폴리펩타이드 D1, D2 등의 선형 조합 b0+b 1D1+b 2D2+...,이다.
| AUC (B vs. V) |
AUC (I vs. NI) |
||||
| Const | IP-10 | TRAIL | 0.88 | 0.91 | |
| δ0 | -0.3333 | 0.0086 | 0.0006 | ||
| δ1 | 2.4481 | 0.0089 | -0.0294 | ||
| Const | CRP | IP-10 | 0.89 | 0.95 | |
| δ0 | -0.474 | 0.0517 | 0.0055 | ||
| δ1 | -1.9201 | 0.0902 | 0.0046 | ||
| Const | IP-10 | SAA | 0.85647 | 0.96324 | |
| δ0 | -1.0634 | 0.0089 | 9623.7195 | ||
| δ1 | -2.0098 | 0.0079 | 14280.3897 | ||
| Const | IL1ra | IP-10 | 0.63901 | 0.89408 | |
| δ0 | -0.2347 | 77589304.64 | 0.0077 | ||
| δ1 | 0.3245 | 122880671.4 | 0.0069 | ||
| Const | IP-10 | PCT | 0.70468 | 0.735 | |
| δ0 | 1.3717 | 0.0012 | 0.1778 | ||
| δ1 | 1.3073 | 0.0007 | 0.9426 | ||
| Const | CRP | TRAIL | 0.94 | 0.93 | |
| δ0 | -0.551 | 0.0647 | 0.0129 | ||
| δ1 | -0.1177 | 0.0953 | -0.0077 | ||
| Const | SAA | TRAIL | 0.90714 | 0.92719 | |
| δ0 | -1.0101 | 15457.6689 | 0.0146 | ||
| δ1 | 0.2736 | 18311.8735 | -0.0081 | ||
| Const | IL1ra | TRAIL | 0.88673 | 0.85523 | |
| δ0 | -0.1638 | 660539652.3 | 0.0118 | ||
| δ1 | 3.3011 | 691029794.9 | -0.0224 | ||
| Const | PCT | TRAIL | 0.86706 | 0.69731 | |
| δ0 | 0.7941 | 0.6699 | 0.0095 | ||
| δ1 | 2.4419 | 1.0871 | -0.0105 | ||
| Const | CRP | SAA | 0.89 | 0.92 | |
| δ0 | 0.9937 | 0.0371 | 7927.9578 | ||
| δ1 | -1.2549 | 0.0704 | 9043.9184 | ||
| Const | CRP | IL1ra | 0.87 | 0.93 | |
| δ0 | 0.9321 | 0.0549 | 357544464 | ||
| δ1 | -0.8849 | 0.0895 | 345095895 | ||
| Const | CRP | PCT | 0.88 | 0.85 | |
| δ0 | 1.225 | 0.0543 | 0.1493 | ||
| δ1 | -1.48 | 0.1052 | 0.71 | ||
| Const | IL1ra | SAA | 0.82529 | 0.9154 | |
| δ0 | 0.9453 | 233885248 | 11965 | ||
| δ1 | -0.6599 | 201037678 | 17194.2625 | ||
| Const | PCT | SAA | 0.78722 | 0.84314 | |
| δ0 | 1.4185 | -0.6192 | 6627 | ||
| δ1 | 0.1417 | 0.2744 | 8964 | ||
| Const | IL1ra | PCT | 0.58647 | 0.82323 | |
| δ0 | 1.3547 | 601268546 | -1.0932 | ||
| δ1 | 0.7175 | 600085479 | 0.7431 |
| AUC (B vs. V) |
AUC (I vs. NI) |
|||||
| Const | CRP | IP-10 | TRAIL | 0.94 | 0.96 | |
| δ0 | -1.0317 | 0.0555 | 0.0053 | 0.005 | ||
| δ1 | -0.6693 | 0.0884 | 0.005 | -0.0143 | ||
| Const | IP-10 | SAA | TRAIL | 0.91 | 0.96 | |
| δ0 | -1.636 | 0.0087 | 9804.469 | 0.0047 | ||
| δ1 | -0.435 | 0.0085 | 12810.9197 | -0.0167 | ||
| Const | IL1ra | IP-10 | TRAIL | 0.89 | 0.90 | |
| δ0 | -0.7474 | 24233992.13 | 0.0072 | 0.0056 | ||
| δ1 | 2.6252 | 57162308.55 | 0.0073 | -0.0282 | ||
| Const | IP-10 | PCT | TRAIL | 0.85 | 0.66 | |
| δ0 | 0.71 | 0.0005 | 0.7463 | 0.008 | ||
| δ1 | 2.1832 | 0.001 | 1.1103 | -0.0136 | ||
| Const | CRP | IP-10 | SAA | 0.91325 | 0.97318 | |
| δ0 | -1.1506 | 0.0389 | 0.0079 | 4964.9078 | ||
| δ1 | -2.7684 | 0.0729 | 0.0069 | 6345.7097 | ||
| Const | CRP | IL1ra | IP-10 | 0.90645 | 0.95695 | |
| δ0 | -0.5109 | 0.0635 | -72572842.54 | 0.0062 | ||
| δ1 | -1.6901 | 0.1025 | -16278785.64 | 0.0046 | ||
| Const | CRP | IP-10 | PCT | 0.88475 | 0.8 | |
| δ0 | 0.1233 | 0.0598 | 0.0016 | 0.1083 | ||
| δ1 | -2.1504 | 0.1081 | 0.0011 | 0.6599 | ||
| Const | IL1ra | IP-10 | SAA | 0.85722 | 0.94944 | |
| δ0 | -0.97 | -141670519.4 | 0.0094 | 9571.3145 | ||
| δ1 | -1.932 | -119518794.5 | 0.008 | 15309.775 | ||
| Const | IP-10 | PCT | SAA | 0.79658 | 0.95635 | |
| δ0 | -0.5085 | 0.0047 | -0.6596 | 6137.1652 | ||
| δ1 | -1.3306 | 0.004 | 0.2775 | 8580.4524 | ||
| Const | IL1ra | IP-10 | PCT | 0.69549 | 0.73737 | |
| δ0 | 1.0752 | 540518195.3 | 0.0005 | -1.1448 | ||
| δ1 | 0.9893 | 578154355.6 | -0.0003 | 0.7431 | ||
| Const | CRP | SAA | TRAIL | 0.93838 | 0.94489 | |
| δ0 | -1.1898 | 0.0419 | 8741.563 | 0.0147 | ||
| δ1 | -0.9205 | 0.0715 | 8922.431 | -0.0045 | ||
| Const | CRP | IL1ra | TRAIL | 0.94316 | 0.92941 | |
| δ0 | -1.1214 | 0.0735 | 142723684.3 | 0.0158 | ||
| δ1 | 0.254 | 0.1005 | 142922206.2 | -0.0124 | ||
| Const | CRP | PCT | TRAIL | 0.91373 | 0.85644 | |
| δ0 | -0.695 | 0.066 | 0.3236 | 0.0132 | ||
| δ1 | -1.7666 | 0.1084 | 0.6114 | 0.0019 | ||
| Const | IL1ra | SAA | TRAIL | 0.91698 | 0.91298 | |
| δ0 | -1.1796 | 19314215.49 | 13897.6693 | 0.0165 | ||
| δ1 | 0.5955 | -373899.4284 | 17471.1789 | -0.0114 | ||
| Const | PCT | SAA | TRAIL | 0.85 | 0.9451 | |
| δ0 | -2.1844 | -0.0348 | 13902.8636 | 0.0281 | ||
| δ1 | -1.6348 | 0.7361 | 15302.3132 | 0.0141 | ||
| Const | IL1ra | PCT | TRAIL | 0.8797 | 0.73737 | |
| δ0 | -0.3418 | 445497461.8 | 1.6517 | 0.0126 | ||
| δ1 | 2.2766 | 638669048.4 | 2.4203 | -0.0203 | ||
| Const | CRP | IL1ra | SAA | 0.88856 | 0.91932 | |
| δ0 | 0.8638 | 0.0265 | 224710899.2 | 7641.7563 | ||
| δ1 | -1.256 | 0.0536 | 201425116.6 | 9730.7248 | ||
| Const | CRP | PCT | SAA | 0.88556 | 0.90588 | |
| δ0 | 1.1579 | 0.0207 | -1.4792 | 8520.704 | ||
| δ1 | -1.3994 | 0.0695 | -0.2234 | 7599.3621 | ||
| Const | CRP | IL1ra | PCT | 0.86842 | 0.84343 | |
| δ0 | 0.8368 | 0.0388 | 547844063.4 | -0.6599 | ||
| δ1 | -1.6604 | 0.0873 | 473174484.1 | -0.1506 | ||
| Const | IL1ra | PCT | SAA | 0.81448 | 0.9 | |
| δ0 | 1.0109 | 565967860.9 | -2.3088 | 10349.4815 | ||
| δ1 | -1.0283 | 515166286.4 | -0.2687 | 15172.8663 |
| AUC (B vs. V) |
AUC (I vs. NI) |
||||||
| Const | CRP | IP-10 | SAA | TRAIL | 0.94 | 0.97 | |
| δ0 | -1.7981 | 0.0394 | 0.0075 | 5383.841 | 0.0058 | ||
| δ1 | -1.5541 | 0.0702 | 0.007 | 5731.9467 | -0.012 | ||
| Const | CRP | IL1ra | IP-10 | TRAIL | 0.94 | 0.96 | |
| δ0 | -1.5229 | 0.0703 | -6.995E+7 | 0.0053 | 0.0091 | ||
| δ1 | -0.2128 | 0.101 | -3.228E+7 | 0.0046 | -0.0166 | ||
| Const | CRP | IP-10 | PCT | TRAIL | 0.903 | 0.78667 | |
| δ0 | -0.6733 | 0.0651 | 0.0007 | 0.2921 | 0.0101 | ||
| δ1 | -1.8383 | 0.1077 | 0.001 | 0.5293 | -0.0021 | ||
| Const | IL1ra | IP-10 | SAA | TRAIL | 0.91777 | 0.94957 | |
| δ0 | -2.036 | -153195983.2 | 0.0088 | 10289.5699 | 0.0091 | ||
| δ1 | -0.2825 | -138993063.2 | 0.0082 | 14282.9357 | -0.0169 | ||
| Const | IP-10 | PCT | SAA | TRAIL | 0.8433 | 0.93254 | |
| δ0 | -3.3387 | 0.0068 | -0.2264 | 12161.0003 | 0.0218 | ||
| δ1 | -2.9001 | 0.0068 | 0.5366 | 13578.3133 | 0.0083 | ||
| Const | IL1ra | IP-10 | PCT | TRAIL | 0.86842 | 0.65657 | |
| δ0 | -0.356 | 481673333.4 | -0.0004 | 1.6805 | 0.0147 | ||
| δ1 | 2.4805 | 491494579.8 | 0.001 | 2.4993 | -0.0268 | ||
| Const | CRP | IL1ra | IP-10 | SAA | 0.92002 | 0.95829 | |
| δ0 | -1.0249 | 0.028 | -1.5446E+8 | 0.0088 | 6131.1692 | ||
| δ1 | -2.3655 | 0.0614 | -9.6352E+7 | 0.0067 | 8579.4749 | ||
| Const | CRP | IP-10 | PCT | SAA | 0.8735 | 0.9881 | |
| δ0 | -1.4913 | 0.0419 | 0.0064 | -1.4641 | 4377.1407 | ||
| δ1 | -3.62 | 0.0857 | 0.0059 | -0.1982 | 3810.7522 | ||
| Const | CRP | IL1ra | IP-10 | PCT | 0.89098 | 0.74242 | |
| δ0 | 0.6379 | 0.0378 | 4.54739E+8 | 0.0004 | -0.4843 | ||
| δ1 | -0.7642 | 0.0969 | 4.84865E+8 | -0.0018 | -0.2044 | ||
| Const | IL1ra | IP-10 | PCT | SAA | 0.77828 | 0.94444 | |
| δ0 | -0.802 | -212692846.6 | 0.0095 | -2.8236 | 4951.1109 | ||
| δ1 | -2.0339 | -210027138.1 | 0.008 | -0.1446 | 10430.5725 | ||
| Const | CRP | IL1ra | SAA | TRAIL | 0.93742 | 0.92564 | |
| δ0 | -1.3076 | 0.0386 | 2.10729E+7 | 8701.5399 | 0.0163 | ||
| δ1 | -0.2694 | 0.062 | 1.31614E+7 | 9890.6956 | -0.0099 | ||
| Const | CRP | PCT | SAA | TRAIL | 0.91111 | 0.95294 | |
| δ0 | -1.8221 | 0.0278 | -1.3285 | 11551.5028 | 0.0253 | ||
| δ1 | -2.7829 | 0.0748 | -0.2648 | 9802.9581 | 0.0141 | ||
| Const | CRP | IL1ra | PCT | TRAIL | 0.89474 | 0.79798 | |
| δ0 | -0.6344 | 0.0476 | 2.756E+8 | -0.1689 | 0.0137 | ||
| δ1 | 0.5587 | 0.0994 | 2.7563E+8 | -0.236 | -0.0264 | ||
| Const | IL1ra | PCT | SAA | TRAIL | 0.92308 | 0.85556 | |
| δ0 | -2.2741 | 432026875.9 | -0.5098 | 12347.4916 | 0.0343 | ||
| δ1 | 0.0383 | 426850211.8 | -0.4124 | 19586.5686 | -0.0152 | ||
| Const | CRP | IL1ra | PCT | SAA | 0.85068 | 0.9 | |
| δ0 | 0.5611 | 0.0552 | 6.42961E+8 | -0.5099 | 2665.2949 | ||
| δ1 | -1.8313 | 0.0941 | 5.88426E+8 | -0.3614 | 3734.4091 |
| AUC (B vs. V) |
AUC (I vs. NI) |
|||||||
| Const | CRP | IL1ra | IP-10 | SAA | TRAIL | 0.94381 | 0.95963 | |
| δ0 | -2.1333 | 0.0336 | -1.6265E+8 | 0.0082 | 6688.18 | 0.0092 | ||
| δ1 | -1.1202 | 0.0619 | -1.17187E+8 | 0.0069 | 8261.93 | -0.0136 | ||
| Const | CRP | IP-10 | PCT | SAA | TRAIL | 0.89972 | 0.95635 | |
| δ0 | -3.4117 | 0.0517 | 0.0074 | -1.297 | 6302.89 | 0.0178 | ||
| δ1 | -4.4957 | 0.1 | 0.0076 | -0.249 | 4437.96 | 0.0063 | ||
| Const | CRP | IL1ra | IP-10 | PCT | TRAIL | 0.88346 | 0.71717 | |
| δ0 | -1.3864 | 0.0616 | 5.4749E+8 | -0.0017 | -0.2302 | 0.0246 | ||
| δ1 | -0.1627 | 0.1096 | 5.0261E+8 | -0.0012 | -0.2819 | -0.017 | ||
| Const | IL1ra | IP-10 | PCT | SAA | TRAIL | 0.87783 | 0.85556 | |
| δ0 | -6.1686 | 2530173.292 | 0.0149 | -0.7334 | 5922.72 | 0.0529 | ||
| δ1 | -3.7073 | 32115407.24 | 0.0139 | -0.282 | 14225.92 | 0.0043 | ||
| Const | CRP | IL1ra | IP-10 | PCT | SAA | 0.819 | 0.91111 | |
| δ0 | -2.8588 | 0.1586 | -8.7081E+8 | 0.0141 | -0.2611 | -22863.96 | ||
| δ1 | -3.2596 | 0.2362 | 7.27742E+8 | 0.008 | -0.3918 | -18573.7 | ||
| Const | CRP | IL1ra | PCT | SAA | TRAIL | 0.90045 | 0.87778 | |
| δ0 | -3.522 | 0.1355 | 6.98426E+8 | -0.4075 | -7661.57 | 0.0397 | ||
| δ1 | -1.7514 | 0.1689 | 6.53495E+8 | -0.4915 | -4178.89 | -0.008 |
실시예
13
비 선형
함수를 포함하는 예시적인 좌표
본 발명자에 의해, 좌표 δ1 및 δ2의 계산에서 비선형 함수 φ0 및 φ1의 결합은 결과의 의미있는 해석을 가능하게 하는 확률적 프레임 워크를 유지하면서 데이터에서 보다 미묘한 경향을 알아낸다는 것을 예상치 못하게 발견하였다. 이 실시예에서, 좌표 δ1 및 δ2은 다음의 방정식에 따라 계산된다 :
δ0 = a 0 + a 1C + a 2I + a 3T + φ0
δ1 = b 0 + b 1C + b 2I + b 3T + φ1,
그리고 상기 비선형 함수는 다음과 같이 정의된다:
φ0 = q 1Cγ1 + q 2Cγ2 + q 3Tγ3
φ1 = r 1Cγ1 + r 2Cγ2 + r 3Tγ3.
상기 γ1=0.5, γ2=2 및 γ3=0.5이다.
표 39은 이 실시예에서 사용된 계수 및 상수를 상세히 설명한다.
| 제 2 좌표 δ1 (박테리아성) |
제 1
좌표
δ 0 (바이러스성) |
|
| Const | b 0 =5.5123 | a 0 =-0.8388 |
| CRP (mg/ml) | b 1 = -0.0636 | a 1 = -0.0487 |
| CRP 0 .5 (mg/ml) 0.5 | r 1 = 1.4877 | q 1 =1.1367 |
| CRP 2 (mg/ml) 2 | r 2 =3.5010-05 | q 2 =-5.1410 -05 |
| IP10 ( pg /ml) | b 2 = 0.0085 | a 2 = 0.0089 |
| TRAIL ( pg /ml) | b 3 = 0.0646 | a 3 = 0.0408 |
| TRAIL 0 .5 ( pg /ml) 0.5 | r 3 =-1.8039 | q 3 =-0.6064 |
표 39에 제시된 모델의 성능은 미생물학적으로 확인된 집단 (AUC는 0.95 ± 0.03), 만장일치 집단 (AUC는 0.95 ± 0.02) 및 연구 집단 (AUC는 0.93 ± 0.02)에서 조사되었다. 불확실한 영역의 크기가 증가함에 따라 시그니쳐의 성능이 향상되었다.
아래 표 40-42는 본 실시예의 비선형 모델을 사용하는 경우 박테리아성 대 바이러스성 감염을 진단하기 위한 시그니쳐 척도의 정확성을 상세히 설명한다. 성능 평가치 및 95 % CI를 표시된 것과 같이 다른 크기의 불확실한 영역을 사용하여 미생물학적으로 확인된 하위 집단 (표 40, n = 241), 만장일치 하위 집단 (표 41, n = 527) 및 연구 집단 (표 42, n = 653)에서 얻었다. 아래 표 43-45는 다른 임계값을 적용할 때 연구 집단에서 불확실한 면역 반응을 보인 환자의 백분율을 나타내며, 표 46-47은 연구 집단에서 얻은 상이한 불확실한 면역 반응 임계값를 적용할 때 시그니쳐의 민감도와 특이성을 상세히 설명한다. 표 43-47에서 가장 왼쪽의 열은 최소 불확실한 면역 반응 임계값을 나타내고, 가장 위쪽의 행은 최대 불확실한 면역 반응 임계값을 나타낸다.
| 정확성 측정 | 모든 환자 | 불확실한 면역 반응 필터 (35-65) | 불확실한 면역 반응 필터 (30-70) | 불확실한 면역 반응 필터 (20-80) | 불확실한 면역 반응 필터 (10-90) |
| 전체 정확성 | 0.89, (0.85, 0.93) |
0.93, (0.90, 0.97) |
0.94, (0.91, 0.97) |
0.96, (0.93, 0.99) |
0.98, (0.96, 1.00) |
| 민감도 | 0.88, (0.80, 0.96) |
0.93, (0.87, 1.00) |
0.95, (0.89, 1.00) |
0.96, (0.91, 1.00) |
0.96, (0.90, 1.00) |
| 특이성 | 0.90, (0.87, 0.94) |
0.94, (0.90, 0.97) |
0.94, (0.90, 0.98) |
0.96, (0.93, 0.99) |
0.99, (0.97, 1.00) |
| 포함된 환자 % | 100% | 90% | 87% | 78% | 65% |
| 정확성 측정 | 모든 환자 | 불확실한 면역 반응 필터 (35-65) | 불확실한 면역 반응 필터 (30-70) | 불확실한 면역 반응 필터 (20-80) | 불확실한 면역 반응 필터 (10-90) |
| 전체 정확성 | 0.88, (0.85, 0.91) |
0.92, (0.89, 0.94) |
0.93, (0.90, 0.95) |
0.95, (0.93, 0.97) |
0.97, (0.95, 0.99) |
| 민감도 | 0.85, (0.81, 0.89) |
0.90, (0.86, 0.94) |
0.91, (0.87, 0.95) |
0.93, (0.90, 0.97) |
0.96, (0.93, 0.99) |
| 특이성 | 0.91, (0.87, 0.94) |
0.93, (0.90, 0.97) |
0.94, (0.91, 0.97) |
0.96, (0.93, 0.99) |
0.98, (0.96, 1.00) |
| 포함된 환자 % | 100% | 90% | 86% | 76% | 63% |
| 정확성 측정 | 모든 환자 | 불확실한 면역 반응 필터 (35-65) | 불확실한 면역 반응 필터 (30-70) | 불확실한 면역 반응 필터 (20-80) | 불확실한 면역 반응 필터 (10-90) |
| 전체 정확성 | 0.85, (0.83, 0.88) |
0.89, (0.86, 0.91) |
0.90, (0.87, 0.92) |
0.92, (0.90, 0.95) |
0.95, (0.93, 0.98) |
| 민감도 | 0.83, (0.79, 0.87) |
0.87, (0.83, 0.91) |
0.89, (0.85, 0.92) |
0.92, (0.88, 0.95) |
0.95, (0.91, 0.98) |
| 특이성 | 0.87, (0.84, 0.91) |
0.90, (0.87, 0.94) |
0.91, (0.88, 0.95) |
0.93, (0.89, 0.96) |
0.95, (0.92, 0.98) |
| 포함된 환자 % | 100% | 88% | 84% | 72% | 58% |
| 0.1 | 0.15 | 0.2 | 0.25 | 0.3 | 0.35 | 0.4 | 0.45 | 0.5 | 0.55 | 0.6 | 0.65 | 0.7 | 0.75 | 0.8 | 0.85 | 0.9 | 0.95 | |
| 0.05 | 6.6 | 10.4 | 13.9 | 17.6 | 20.5 | 22.7 | 24.0 | 26.3 | 29.1 | 31.2 | 33.2 | 34.8 | 36.3 | 38.6 | 40.9 | 44.0 | 47.2 | 52.8 |
| 0.1 | 3.8 | 7.4 | 11.0 | 13.9 | 16.1 | 17.5 | 19.8 | 22.5 | 24.7 | 26.6 | 28.2 | 29.7 | 32.0 | 34.3 | 37.4 | 40.6 | 46.2 | |
| 0.15 | 3.5 | 7.2 | 10.1 | 12.3 | 13.6 | 15.9 | 18.7 | 20.8 | 22.8 | 24.3 | 25.9 | 28.2 | 30.5 | 33.5 | 36.8 | 42.4 | ||
| 0.2 | 3.7 | 6.6 | 8.7 | 10.1 | 12.4 | 15.2 | 17.3 | 19.3 | 20.8 | 22.4 | 24.7 | 27.0 | 30.0 | 33.2 | 38.9 | |||
| 0.25 | 2.9 | 5.1 | 6.4 | 8.7 | 11.5 | 13.6 | 15.6 | 17.2 | 18.7 | 21.0 | 23.3 | 26.3 | 29.6 | 35.2 | ||||
| 0.3 | 2.1 | 3.5 | 5.8 | 8.6 | 10.7 | 12.7 | 14.2 | 15.8 | 18.1 | 20.4 | 23.4 | 26.6 | 32.3 | |||||
| 0.35 | 1.4 | 3.7 | 6.4 | 8.6 | 10.6 | 12.1 | 13.6 | 15.9 | 18.2 | 21.3 | 24.5 | 30.2 | ||||||
| 0.4 | 2.3 | 5.1 | 7.2 | 9.2 | 10.7 | 12.3 | 14.5 | 16.8 | 19.9 | 23.1 | 28.8 | |||||||
| 0.45 | 2.8 | 4.9 | 6.9 | 8.4 | 10.0 | 12.3 | 14.5 | 17.6 | 20.8 | 26.5 | ||||||||
| 0.5 | 2.1 | 4.1 | 5.7 | 7.2 | 9.5 | 11.8 | 14.9 | 18.1 | 23.7 | |||||||||
| 0.55 | 2.0 | 3.5 | 5.1 | 7.4 | 9.6 | 12.7 | 15.9 | 21.6 | ||||||||||
| 0.6 | 1.5 | 3.1 | 5.4 | 7.7 | 10.7 | 13.9 | 19.6 | |||||||||||
| 0.65 | 1.5 | 3.8 | 6.1 | 9.2 | 12.4 | 18.1 | ||||||||||||
| 0.7 | 2.3 | 4.6 | 7.7 | 10.9 | 16.5 | |||||||||||||
| 0.75 | 2.3 | 5.4 | 8.6 | 14.2 | ||||||||||||||
| 0.8 | 3.1 | 6.3 | 11.9 | |||||||||||||||
| 0.85 | 3.2 | 8.9 | ||||||||||||||||
| 0.9 | 5.7 |
| 0.1 | 0.15 | 0.2 | 0.25 | 0.3 | 0.35 | 0.4 | 0.45 | 0.5 | 0.55 | 0.6 | 0.65 | 0.7 | 0.75 | 0.8 | 0.85 | 0.9 | 0.95 | |
| 0.05 | 2.5 | 3.4 | 5.3 | 7.5 | 9.1 | 10.3 | 11.3 | 13.8 | 16.6 | 18.8 | 21.6 | 23.5 | 26.0 | 29.5 | 33.5 | 38.2 | 43.6 | 53.6 |
| 0.1 | 0.9 | 2.8 | 5.0 | 6.6 | 7.8 | 8.8 | 11.3 | 14.1 | 16.3 | 19.1 | 21.0 | 23.5 | 27.0 | 31.0 | 35.7 | 41.1 | 51.1 | |
| 0.15 | 1.9 | 4.1 | 5.6 | 6.9 | 7.8 | 10.3 | 13.2 | 15.4 | 18.2 | 20.1 | 22.6 | 26.0 | 30.1 | 34.8 | 40.1 | 50.2 | ||
| 0.2 | 2.2 | 3.8 | 5.0 | 6.0 | 8.5 | 11.3 | 13.5 | 16.3 | 18.2 | 20.7 | 24.1 | 28.2 | 32.9 | 38.2 | 48.3 | |||
| 0.25 | 1.6 | 2.8 | 3.8 | 6.3 | 9.1 | 11.3 | 14.1 | 16.0 | 18.5 | 21.9 | 26.0 | 30.7 | 36.1 | 46.1 | ||||
| 0.3 | 1.3 | 2.2 | 4.7 | 7.5 | 9.7 | 12.5 | 14.4 | 16.9 | 20.4 | 24.5 | 29.2 | 34.5 | 44.5 | |||||
| 0.35 | 0.9 | 3.4 | 6.3 | 8.5 | 11.3 | 13.2 | 15.7 | 19.1 | 23.2 | 27.9 | 33.2 | 43.3 | ||||||
| 0.4 | 2.5 | 5.3 | 7.5 | 10.3 | 12.2 | 14.7 | 18.2 | 22.3 | 27.0 | 32.3 | 42.3 | |||||||
| 0.45 | 2.8 | 5.0 | 7.8 | 9.7 | 12.2 | 15.7 | 19.7 | 24.5 | 29.8 | 39.8 | ||||||||
| 0.5 | 2.2 | 5.0 | 6.9 | 9.4 | 12.9 | 16.9 | 21.6 | 27.0 | 37.0 | |||||||||
| 0.55 | 2.8 | 4.7 | 7.2 | 10.7 | 14.7 | 19.4 | 24.8 | 34.8 | ||||||||||
| 0.6 | 1.9 | 4.4 | 7.8 | 11.9 | 16.6 | 21.9 | 32.0 | |||||||||||
| 0.65 | 2.5 | 6.0 | 10.0 | 14.7 | 20.1 | 30.1 | ||||||||||||
| 0.7 | 3.4 | 7.5 | 12.2 | 17.6 | 27.6 | |||||||||||||
| 0.75 | 4.1 | 8.8 | 14.1 | 24.1 | ||||||||||||||
| 0.8 | 4.7 | 10.0 | 20.1 | |||||||||||||||
| 0.85 | 5.3 | 15.4 | ||||||||||||||||
| 0.9 | 10.0 |
| 0.1 | 0.15 | 0.2 | 0.25 | 0.3 | 0.35 | 0.4 | 0.45 | 0.5 | 0.55 | 0.6 | 0.65 | 0.7 | 0.75 | 0.8 | 0.85 | 0.9 | 0.95 | |
| 0.05 | 10.5 | 17.1 | 22.2 | 27.2 | 31.4 | 34.4 | 36.2 | 38.3 | 41.0 | 43.1 | 44.3 | 45.5 | 46.1 | 47.3 | 47.9 | 49.4 | 50.6 | 52.1 |
| 0.1 | 6.6 | 11.7 | 16.8 | 21.0 | 24.0 | 25.7 | 27.8 | 30.5 | 32.6 | 33.8 | 35.0 | 35.6 | 36.8 | 37.4 | 38.9 | 40.1 | 41.6 | |
| 0.15 | 5.1 | 10.2 | 14.4 | 17.4 | 19.2 | 21.3 | 24.0 | 26.0 | 27.2 | 28.4 | 29.0 | 30.2 | 30.8 | 32.3 | 33.5 | 35.0 | ||
| 0.2 | 5.1 | 9.3 | 12.3 | 14.1 | 16.2 | 18.9 | 21.0 | 22.2 | 23.4 | 24.0 | 25.1 | 25.7 | 27.2 | 28.4 | 29.9 | |||
| 0.25 | 4.2 | 7.2 | 9.0 | 11.1 | 13.8 | 15.9 | 17.1 | 18.3 | 18.9 | 20.1 | 20.7 | 22.2 | 23.4 | 24.9 | ||||
| 0.3 | 3.0 | 4.8 | 6.9 | 9.6 | 11.7 | 12.9 | 14.1 | 14.7 | 15.9 | 16.5 | 18.0 | 19.2 | 20.7 | |||||
| 0.35 | 1.8 | 3.9 | 6.6 | 8.7 | 9.9 | 11.1 | 11.7 | 12.9 | 13.5 | 15.0 | 16.2 | 17.7 | ||||||
| 0.4 | 2.1 | 4.8 | 6.9 | 8.1 | 9.3 | 9.9 | 11.1 | 11.7 | 13.2 | 14.4 | 15.9 | |||||||
| 0.45 | 2.7 | 4.8 | 6.0 | 7.2 | 7.8 | 9.0 | 9.6 | 11.1 | 12.3 | 13.8 | ||||||||
| 0.5 | 2.1 | 3.3 | 4.5 | 5.1 | 6.3 | 6.9 | 8.4 | 9.6 | 11.1 | |||||||||
| 0.55 | 1.2 | 2.4 | 3.0 | 4.2 | 4.8 | 6.3 | 7.5 | 9.0 | ||||||||||
| 0.6 | 1.2 | 1.8 | 3.0 | 3.6 | 5.1 | 6.3 | 7.8 | |||||||||||
| 0.65 | 0.6 | 1.8 | 2.4 | 3.9 | 5.1 | 6.6 | ||||||||||||
| 0.7 | 1.2 | 1.8 | 3.3 | 4.5 | 6.0 | |||||||||||||
| 0.75 | 0.6 | 2.1 | 3.3 | 4.8 | ||||||||||||||
| 0.8 | 1.5 | 2.7 | 4.2 | |||||||||||||||
| 0.85 | 1.2 | 2.7 | ||||||||||||||||
| 0.9 | 1.5 |
| 0.1 | 0.15 | 0.2 | 0.25 | 0.3 | 0.35 | 0.4 | 0.45 | 0.5 | 0.55 | 0.6 | 0.65 | 0.7 | 0.75 | 0.8 | 0.85 | 0.9 | 0.95 | |
| 0.05 | 84.6 | 85.4 | 87.1 | 89.2 | 90.7 | 92.0 | 92.9 | 95.6 | 98.9 | 98.8 | 98.8 | 98.8 | 98.7 | 98.7 | 98.6 | 98.5 | 98.3 | 98.0 |
| 0.1 | 83.2 | 84.8 | 86.8 | 88.3 | 89.5 | 90.4 | 92.9 | 96.0 | 95.9 | 95.7 | 95.6 | 95.5 | 95.3 | 95.0 | 94.6 | 94.1 | 92.9 | |
| 0.15 | 84.0 | 85.9 | 87.4 | 88.6 | 89.5 | 92.0 | 94.9 | 94.8 | 94.6 | 94.5 | 94.3 | 94.1 | 93.7 | 93.3 | 92.7 | 91.2 | ||
| 0.2 | 84.3 | 85.7 | 86.8 | 87.7 | 90.1 | 92.9 | 92.8 | 92.5 | 92.3 | 92.1 | 91.7 | 91.3 | 90.7 | 89.8 | 87.9 | |||
| 0.25 | 83.8 | 84.8 | 85.7 | 88.0 | 90.7 | 90.5 | 90.1 | 89.9 | 89.6 | 89.2 | 88.6 | 87.8 | 86.8 | 84.3 | ||||
| 0.3 | 83.5 | 84.3 | 86.5 | 89.2 | 88.9 | 88.5 | 88.3 | 87.9 | 87.4 | 86.7 | 85.8 | 84.7 | 81.9 | |||||
| 0.35 | 83.2 | 85.4 | 88.0 | 87.7 | 87.3 | 87.0 | 86.6 | 86.0 | 85.3 | 84.3 | 83.1 | 80.1 | ||||||
| 0.4 | 84.6 | 87.1 | 86.8 | 86.4 | 86.1 | 85.7 | 85.1 | 84.3 | 83.3 | 81.9 | 78.8 | |||||||
| 0.45 | 84.8 | 84.5 | 84.0 | 83.7 | 83.2 | 82.5 | 81.6 | 80.5 | 79.0 | 75.5 | ||||||||
| 0.5 | 82.1 | 81.5 | 81.1 | 80.6 | 79.9 | 78.9 | 77.6 | 76.0 | 72.1 | |||||||||
| 0.55 | 81.9 | 81.6 | 81.1 | 80.4 | 79.4 | 78.2 | 76.7 | 73.1 | ||||||||||
| 0.6 | 82.1 | 81.6 | 81.0 | 80.1 | 78.9 | 77.5 | 74.2 | |||||||||||
| 0.65 | 82.0 | 81.3 | 80.5 | 79.4 | 78.0 | 74.9 | ||||||||||||
| 0.7 | 81.8 | 81.0 | 80.0 | 78.7 | 75.8 | |||||||||||||
| 0.75 | 81.7 | 80.8 | 79.6 | 76.9 | ||||||||||||||
| 0.8 | 81.6 | 80.5 | 78.0 | |||||||||||||||
| 0.85 | 81.5 | 79.3 | ||||||||||||||||
| 0.9 | 80.5 |
| 0.1 | 0.15 | 0.2 | 0.25 | 0.3 | 0.35 | 0.4 | 0.45 | 0.5 | 0.55 | 0.6 | 0.65 | 0.7 | 0.75 | 0.8 | 0.85 | 0.9 | 0.95 | |
| 0.05 | 86.3 | 85.2 | 84.2 | 83.1 | 82.1 | 81.3 | 80.8 | 80.1 | 79.2 | 82.1 | 83.9 | 85.7 | 86.7 | 88.6 | 89.7 | 92.3 | 94.5 | 97.5 |
| 0.1 | 86.9 | 86.1 | 85.3 | 84.5 | 83.9 | 83.5 | 83.0 | 82.3 | 84.9 | 86.4 | 88.0 | 88.8 | 90.5 | 91.4 | 93.6 | 95.5 | 97.9 | |
| 0.15 | 87.1 | 86.3 | 85.7 | 85.1 | 84.8 | 84.4 | 83.9 | 86.2 | 87.7 | 89.1 | 89.9 | 91.4 | 92.2 | 94.2 | 95.9 | 98.2 | ||
| 0.2 | 87.1 | 86.5 | 86.0 | 85.7 | 85.4 | 84.9 | 87.1 | 88.5 | 89.8 | 90.6 | 92.0 | 92.7 | 94.7 | 96.2 | 98.3 | |||
| 0.25 | 87.2 | 86.8 | 86.5 | 86.2 | 85.8 | 87.9 | 89.2 | 90.5 | 91.1 | 92.5 | 93.2 | 95.0 | 96.5 | 98.4 | ||||
| 0.3 | 87.3 | 87.1 | 86.8 | 86.4 | 88.5 | 89.7 | 90.9 | 91.6 | 92.9 | 93.5 | 95.3 | 96.7 | 98.5 | |||||
| 0.35 | 87.5 | 87.2 | 86.9 | 88.9 | 90.0 | 91.2 | 91.9 | 93.1 | 93.8 | 95.4 | 96.8 | 98.5 | ||||||
| 0.4 | 87.5 | 87.1 | 89.1 | 90.2 | 91.4 | 92.0 | 93.3 | 93.9 | 95.5 | 96.9 | 98.6 | |||||||
| 0.45 | 87.4 | 89.3 | 90.4 | 91.6 | 92.2 | 93.4 | 94.0 | 95.6 | 96.9 | 98.6 | ||||||||
| 0.5 | 89.6 | 90.7 | 91.8 | 92.4 | 93.6 | 94.2 | 95.8 | 97.0 | 98.7 | |||||||||
| 0.55 | 88.8 | 89.9 | 90.4 | 91.6 | 92.1 | 93.6 | 94.8 | 96.4 | ||||||||||
| 0.6 | 88.8 | 89.3 | 90.4 | 91.0 | 92.4 | 93.6 | 95.1 | |||||||||||
| 0.65 | 88.3 | 89.3 | 89.9 | 91.3 | 92.4 | 93.9 | ||||||||||||
| 0.7 | 88.8 | 89.3 | 90.7 | 91.8 | 93.3 | |||||||||||||
| 0.75 | 88.3 | 89.6 | 90.7 | 92.1 | ||||||||||||||
| 0.8 | 89.1 | 90.2 | 91.6 | |||||||||||||||
| 0.85 | 88.8 | 90.2 | ||||||||||||||||
| 0.9 | 89.1 |
상기 시그니쳐 성능을 다음 두 서브 그룹을 제외하고 연구 집단에서 더 조사하였다 : (i) 병원에서 3 일 이상의 항생제 치료 후 혈액 샘플을 채취한 환자 및 (ii) 위장염이 의심되는 환자. 미생물학적으로 확인된 집단 (0.96 ± 0.04의 AUC), 만장일치 집단 (0.96 ± 0.02의 AUC) 및 연구 집단 (0.95 ± 0.02의 AUC)에 대한 모델 성능의 세부 사항이 표 48-50에 추가로 기술되어 있다.
표 48-50은 비선형 MLR 모델을 사용하여 박테리아성 대 바이러스성 감염을 진단하기 위한 시그니쳐 척도의 정확성을 상세히 설명한다. 병원에서 항생제 치료 3 일 이상 받은 환자 및/또는 위장염 의심 환자를 제외할 때, 성능 평가치 및 95 % CI를 미생물학적으로 확인된 하위 집단 (표 48, n = 200), 만장일치 하위 집단 (표 49, n = 402) 및 연구 집단 (표 50, n = 491)에서 얻었다.
| 정확성 측정 | 모든 환자 | 불확실한 면역 반응 필터 (35-65) | 불확실한 면역 반응 필터 (30-70) | 불확실한 면역 반응 필터 (20-80) | 불확실한 면역 반응 필터 (10-90) |
| 전체 정확성 | 0.91, (0.86, 0.95) |
0.95, (0.92, 1) |
0.95, (0.92, 0.99) |
0.96, (0.93, 0.99) |
0.98, (0.96, 1) |
| 민감도 | 0.90, (0.82, 0.99) |
0.96, (0.89, 1) |
0.96, (0.89, 1) |
0.95, (0.89, 1) |
0.94, (87, 1) |
| 특이성 | 0.91, (0.86, 0.95) |
0.95, (0.91, 0.99) |
0.95, (0.92, 0.99) |
0.97, (0.93, 1) |
1, (1,1) |
| 포함된 환자 % | 100% | 90% | 88% | 80% | 65% |
| 정확성 측정 | 모든 환자 | 불확실한 면역 반응 필터 (35-65) | 불확실한 면역 반응 필터 (30-70) | 불확실한 면역 반응 필터 (20-80) | 불확실한 면역 반응 필터 (10-90) |
| 전체 정확성 | 0.91, (0.88, 0.94) |
0.94, (0.92, 0.97) |
0.95, (0.93, 0.97) |
0.96, (0.94, 0.98) |
0.98, (0.96, 1) |
| 민감도 | 0.89, (0.85, 0.94) |
0.93, (0.89, 0.97) |
0.94, (0.90, 0.98) |
0.95, (0.92, 0.99) |
0.98, (0.95,1) |
| 특이성 | 0.92, (0.88, 0.96) |
0.95, (0.92, 0.98) |
0.95, (0.93, 0.98) |
0.97, (0.94, 0.99) |
0.99, (0.97, 1) |
| 포함된 환자 % | 100% | 91% | 88% | 79% | 65% |
| 정확성 측정 | 모든 환자 | 불확실한 면역 반응 필터 (35-65) |
불확실한 면역 반응 필터 (30-70) |
불확실한 면역 반응 필터 (20-80) |
불확실한 면역 반응 필터 (10-90) |
| 전체 정확성 | 0.88, (0.85, 0.91) |
0.91, (0.89, 0.94) |
0.93, (0.90, 0.95) |
0.94, (0.92, 0.97) |
0.97, (0.95, 0.99) |
| 민감도 | 0.87, (0.83, 0.92) |
0.91, (0.87, 0.95) |
0.92, (0.88, 0.96) |
0.95, (0.91, 0.98) |
0.97, (0.94, 1) |
| 특이성 | 0.89, (0.85, 0.92) |
0.92, (0.89, 0.96) |
0.93, (0.90, 0.96) |
0.94, (0.91, 0.97) |
0.97, (0.94, 1) |
| 포함된 환자 % | 100% | 88% | 85% | 74% | 59% |
실시예
14
항생제 기반 계층화
급성 감염의 의심이 있는 653 명의 환자 중 427 명이 항생제 (299 건의 박테리아성 진단 및 128 건의 바이러스성 진단)를 받았다. 항생제 치료 환자에서 박테리아성 감염자 및 바이러스성 감염 환자를 구별하기 위한 시그니쳐의 AUC는 0.93 ± 0.02였다. 항생제 치료 환자 및 일반 집단의 결과간에 통계적으로 유의한 차이는 없었다 (0.94 ± 0.02 대 0.93 ± 0.02; P = 0.5).
본 발명은 특정 실시예와 관련하여 설명되었지만, 많은 대안, 수정 및 변형이 통상의 기술자에게 용이하다는 것이 명백하다. 따라서, 첨부된 청구 범위의 사상 및 넓은 범위 내에 있는 그러한 모든 대안, 수정 및 변형을 포함한다.
본 명세서에 언급된 모든 간행물, 특허 및 특허 출원은 각각의 개별 간행물, 특허 또는 특허 출원이 구체적으로 및 개별적으로 본 명세서에 참고로 인용된 것과 동등한 정도로 명세서 전체가 본 명세서에 통합된다. 또한, 본 명세서에서 임의의 참조 문헌의 인용 또는 확인은 그러한 참조가 본 발명의 선행 기술로서 이용 가능함을 인정하는 것으로 해석되어서는 안된다. 섹션 표제가 사용되는 한, 반드시 제한적인 것으로 해석되어서는 안된다.
SEQUENCE LISTING
<110> MEMED DIAGNOSTICS LTD.
EDEN, Eran
OVED, Kfir
NAVON, Roy
COHEN-DOTAN, Assaf
BOICO, Olga
<120> COMPUTATIONAL ANALYSIS OF BIOLOGICAL DATA USING MANIFOLD AND A
HYPERPLANE
<130> 62853
<150> US 62/037,180
<151> 2014-08-14
<150> US 62/105,938
<151> 2015-01-21
<160> 38
<170> PatentIn version 3.5
<210> 1
<211> 1953
<212> DNA
<213> Homo sapiens
<400> 1
acagaaccca gaaaaacaac tcattcgctt tcatttcctc actgactata aaagaataga 60
gaaggaaggg cttcagtgac cggctgcctg gctgacttac agcagtcaga ctctgacagg 120
atcatggcta tgatggaggt ccagggggga cccagcctgg gacagacctg cgtgctgatc 180
gtgatcttca cagtgctcct gcagtctctc tgtgtggctg taacttacgt gtactttacc 240
aacgagctga agcagatgca ggacaagtac tccaaaagtg gcattgcttg tttcttaaaa 300
gaagatgaca gttattggga ccccaatgac gaagagagta tgaacagccc ctgctggcaa 360
gtcaagtggc aactccgtca gctcgttaga aagatgattt tgagaacctc tgaggaaacc 420
atttctacag ttcaagaaaa gcaacaaaat atttctcccc tagtgagaga aagaggtcct 480
cagagagtag cagctcacat aactgggacc agaggaagaa gcaacacatt gtcttctcca 540
aactccaaga atgaaaaggc tctgggccgc aaaataaact cctgggaatc atcaaggagt 600
gggcattcat tcctgagcaa cttgcacttg aggaatggtg aactggtcat ccatgaaaaa 660
gggttttact acatctattc ccaaacatac tttcgatttc aggaggaaat aaaagaaaac 720
acaaagaacg acaaacaaat ggtccaatat atttacaaat acacaagtta tcctgaccct 780
atattgttga tgaaaagtgc tagaaatagt tgttggtcta aagatgcaga atatggactc 840
tattccatct atcaaggggg aatatttgag cttaaggaaa atgacagaat ttttgtttct 900
gtaacaaatg agcacttgat agacatggac catgaagcca gtttttttgg ggccttttta 960
gttggctaac tgacctggaa agaaaaagca ataacctcaa agtgactatt cagttttcag 1020
gatgatacac tatgaagatg tttcaaaaaa tctgaccaaa acaaacaaac agaaaacaga 1080
aaacaaaaaa acctctatgc aatctgagta gagcagccac aaccaaaaaa ttctacaaca 1140
cacactgttc tgaaagtgac tcacttatcc caagagaatg aaattgctga aagatctttc 1200
aggactctac ctcatatcag tttgctagca gaaatctaga agactgtcag cttccaaaca 1260
ttaatgcaat ggttaacatc ttctgtcttt ataatctact ccttgtaaag actgtagaag 1320
aaagagcaac aatccatctc tcaagtagtg tatcacagta gtagcctcca ggtttcctta 1380
agggacaaca tccttaagtc aaaagagaga agaggcacca ctaaaagatc gcagtttgcc 1440
tggtgcagtg gctcacacct gtaatcccaa cattttggga acccaaggtg ggtagatcac 1500
gagatcaaga gatcaagacc atagtgacca acatagtgaa accccatctc tactgaaagt 1560
acaaaaatta gctgggtgtg ttggcacatg cctgtagtcc cagctacttg agaggctgag 1620
gcaagagaat tgtttgaacc cgggaggcag aggttgcagt gtggtgagat catgccacta 1680
cactccagcc tggcgacaga gcgagacttg gtttcaaaaa aaaaaaaaaa aaaaacttca 1740
gtaagtacgt gttatttttt tcaataaaat tctattacag tatgtcatgt ttgctgtagt 1800
gctcatattt attgttgttt ttgttttagt actcacttgt ttcataatat caagattact 1860
aaaaatgggg gaaaagactt ctaatctttt tttcataata tctttgacac atattacaga 1920
agaaataaat ttcttacttt taatttaata tga 1953
<210> 2
<211> 1805
<212> DNA
<213> Homo sapiens
<400> 2
acagaaccca gaaaaacaac tcattcgctt tcatttcctc actgactata aaagaataga 60
gaaggaaggg cttcagtgac cggctgcctg gctgacttac agcagtcaga ctctgacagg 120
atcatggcta tgatggaggt ccagggggga cccagcctgg gacagacctg cgtgctgatc 180
gtgatcttca cagtgctcct gcagtctctc tgtgtggctg taacttacgt gtactttacc 240
aacgagctga agcagatgca ggacaagtac tccaaaagtg gcattgcttg tttcttaaaa 300
gaagatgaca gttattggga ccccaatgac gaagagagta tgaacagccc ctgctggcaa 360
gtcaagtggc aactccgtca gctcgttaga aagactccaa gaatgaaaag gctctgggcc 420
gcaaaataaa ctcctgggaa tcatcaagga gtgggcattc attcctgagc aacttgcact 480
tgaggaatgg tgaactggtc atccatgaaa aagggtttta ctacatctat tcccaaacat 540
actttcgatt tcaggaggaa ataaaagaaa acacaaagaa cgacaaacaa atggtccaat 600
atatttacaa atacacaagt tatcctgacc ctatattgtt gatgaaaagt gctagaaata 660
gttgttggtc taaagatgca gaatatggac tctattccat ctatcaaggg ggaatatttg 720
agcttaagga aaatgacaga atttttgttt ctgtaacaaa tgagcacttg atagacatgg 780
accatgaagc cagttttttt ggggcctttt tagttggcta actgacctgg aaagaaaaag 840
caataacctc aaagtgacta ttcagttttc aggatgatac actatgaaga tgtttcaaaa 900
aatctgacca aaacaaacaa acagaaaaca gaaaacaaaa aaacctctat gcaatctgag 960
tagagcagcc acaaccaaaa aattctacaa cacacactgt tctgaaagtg actcacttat 1020
cccaagagaa tgaaattgct gaaagatctt tcaggactct acctcatatc agtttgctag 1080
cagaaatcta gaagactgtc agcttccaaa cattaatgca atggttaaca tcttctgtct 1140
ttataatcta ctccttgtaa agactgtaga agaaagagca acaatccatc tctcaagtag 1200
tgtatcacag tagtagcctc caggtttcct taagggacaa catccttaag tcaaaagaga 1260
gaagaggcac cactaaaaga tcgcagtttg cctggtgcag tggctcacac ctgtaatccc 1320
aacattttgg gaacccaagg tgggtagatc acgagatcaa gagatcaaga ccatagtgac 1380
caacatagtg aaaccccatc tctactgaaa gtacaaaaat tagctgggtg tgttggcaca 1440
tgcctgtagt cccagctact tgagaggctg aggcaagaga attgtttgaa cccgggaggc 1500
agaggttgca gtgtggtgag atcatgccac tacactccag cctggcgaca gagcgagact 1560
tggtttcaaa aaaaaaaaaa aaaaaaactt cagtaagtac gtgttatttt tttcaataaa 1620
attctattac agtatgtcat gtttgctgta gtgctcatat ttattgttgt ttttgtttta 1680
gtactcactt gtttcataat atcaagatta ctaaaaatgg gggaaaagac ttctaatctt 1740
tttttcataa tatctttgac acatattaca gaagaaataa atttcttact tttaatttaa 1800
tatga 1805
<210> 3
<211> 1334
<212> DNA
<213> Homo sapiens
<400> 3
atttcctcac tgactataaa agaatagaga aggaagggct tcagtgaccg gctgcctggc 60
tgacttacag cagtcagact ctgacaggat catggctatg atggaggtcc aggggggacc 120
cagcctggga cagacctgcg tgctgatcgt gatcttcaca gtgctcctgc agtctctctg 180
tgtggctgta acttacgtgt actttaccaa cgagctgaag cagtttgcag aaaatgattg 240
ccagagacta atgtctgggc agcagacagg gtcattgctg ccatcttgaa gtctaccttg 300
ctgagtctac cctgctgacc tcaagcccca tcaaggactg gttgaccctg gcctagacaa 360
ccaccgtgtt tgtaacagca ccaagagcag tcaccatgga aatccacttt tcagaaccaa 420
gggcttctgg agctgaagaa caggcaccca gtgcaagagc tttcttttca gaggcacgca 480
aatgaaaata atccccacac gctaccttct gcccccaatg cccaagtgtg gttagttaga 540
gaatatagcc tcagcctatg atatgctgca ggaaactcat attttgaagt ggaaaggatg 600
ggaggaggcg ggggagacgt atcgtattaa ttatcattct tggaataacc acagcacctc 660
acgtcaaccc gccatgtgtc tagtcaccag cattggccaa gttctatagg agaaactacc 720
aaaattcatg atgcaagaaa catgtgaggg tggagagagt gactggggct tcctctctgg 780
atttctattg ttcagaaatc aatatttatg cataaaaagg tctagaaaga gaaacaccaa 840
aatgacaatg tgatctctag atggtatgat tatgggtact ttttttcctt tttatttttc 900
tatattttac aaattttcta cagggaatgt tataaaaata tccatgctat ccatgtataa 960
ttttcataca gatttaaaga acacagcatt tttatatagt cttatgagaa aacaaccata 1020
ctcaaaatta tgcacacaca cagtctgatc tcacccctgt aaacaagaga tatcatccaa 1080
aggttaagta ggaggtgaga atatagctgc tattagtggt tgttttgttt tgtttttgtg 1140
atttacttat ttagtttttg gagggttttt tttttctttt agaaaagtgt tctttacttt 1200
tccatgcttc cctgcttgcc tgtgtatcct gaatgtatcc aggctttata aactcctggg 1260
taataatgta gctacattaa cttgttaacc tcccatccac ttatacccag gaccttactc 1320
aattttccag gttc 1334
<210> 4
<211> 281
<212> PRT
<213> Homo sapiens
<400> 4
Met Ala Met Met Glu Val Gln Gly Gly Pro Ser Leu Gly Gln Thr Cys
1 5 10 15
Val Leu Ile Val Ile Phe Thr Val Leu Leu Gln Ser Leu Cys Val Ala
20 25 30
Val Thr Tyr Val Tyr Phe Thr Asn Glu Leu Lys Gln Met Gln Asp Lys
35 40 45
Tyr Ser Lys Ser Gly Ile Ala Cys Phe Leu Lys Glu Asp Asp Ser Tyr
50 55 60
Trp Asp Pro Asn Asp Glu Glu Ser Met Asn Ser Pro Cys Trp Gln Val
65 70 75 80
Lys Trp Gln Leu Arg Gln Leu Val Arg Lys Met Ile Leu Arg Thr Ser
85 90 95
Glu Glu Thr Ile Ser Thr Val Gln Glu Lys Gln Gln Asn Ile Ser Pro
100 105 110
Leu Val Arg Glu Arg Gly Pro Gln Arg Val Ala Ala His Ile Thr Gly
115 120 125
Thr Arg Gly Arg Ser Asn Thr Leu Ser Ser Pro Asn Ser Lys Asn Glu
130 135 140
Lys Ala Leu Gly Arg Lys Ile Asn Ser Trp Glu Ser Ser Arg Ser Gly
145 150 155 160
His Ser Phe Leu Ser Asn Leu His Leu Arg Asn Gly Glu Leu Val Ile
165 170 175
His Glu Lys Gly Phe Tyr Tyr Ile Tyr Ser Gln Thr Tyr Phe Arg Phe
180 185 190
Gln Glu Glu Ile Lys Glu Asn Thr Lys Asn Asp Lys Gln Met Val Gln
195 200 205
Tyr Ile Tyr Lys Tyr Thr Ser Tyr Pro Asp Pro Ile Leu Leu Met Lys
210 215 220
Ser Ala Arg Asn Ser Cys Trp Ser Lys Asp Ala Glu Tyr Gly Leu Tyr
225 230 235 240
Ser Ile Tyr Gln Gly Gly Ile Phe Glu Leu Lys Glu Asn Asp Arg Ile
245 250 255
Phe Val Ser Val Thr Asn Glu His Leu Ile Asp Met Asp His Glu Ala
260 265 270
Ser Phe Phe Gly Ala Phe Leu Val Gly
275 280
<210> 5
<211> 101
<212> PRT
<213> Homo sapiens
<400> 5
Met Ala Met Met Glu Val Gln Gly Gly Pro Ser Leu Gly Gln Thr Cys
1 5 10 15
Val Leu Ile Val Ile Phe Thr Val Leu Leu Gln Ser Leu Cys Val Ala
20 25 30
Val Thr Tyr Val Tyr Phe Thr Asn Glu Leu Lys Gln Met Gln Asp Lys
35 40 45
Tyr Ser Lys Ser Gly Ile Ala Cys Phe Leu Lys Glu Asp Asp Ser Tyr
50 55 60
Trp Asp Pro Asn Asp Glu Glu Ser Met Asn Ser Pro Cys Trp Gln Val
65 70 75 80
Lys Trp Gln Leu Arg Gln Leu Val Arg Lys Thr Pro Arg Met Lys Arg
85 90 95
Leu Trp Ala Ala Lys
100
<210> 6
<211> 65
<212> PRT
<213> Homo sapiens
<400> 6
Met Ala Met Met Glu Val Gln Gly Gly Pro Ser Leu Gly Gln Thr Cys
1 5 10 15
Val Leu Ile Val Ile Phe Thr Val Leu Leu Gln Ser Leu Cys Val Ala
20 25 30
Val Thr Tyr Val Tyr Phe Thr Asn Glu Leu Lys Gln Phe Ala Glu Asn
35 40 45
Asp Cys Gln Arg Leu Met Ser Gly Gln Gln Thr Gly Ser Leu Leu Pro
50 55 60
Ser
65
<210> 7
<211> 223
<212> PRT
<213> Homo sapiens
<400> 7
Gln Asp His Gly Tyr Asp Gly Gly Pro Gly Gly Thr Gln Pro Gly Thr
1 5 10 15
Asp Leu Arg Ala Asp Arg Asp Leu His Ser Ala Pro Ala Val Ser Leu
20 25 30
Cys Gly Cys Asn Leu Arg Val Leu Tyr Gln Arg Ala Glu Ala Glu Lys
35 40 45
Gln Gln Asn Ile Ser Pro Leu Val Arg Glu Arg Gly Pro Gln Arg Val
50 55 60
Ala Ala His Ile Thr Gly Thr Arg Gly Arg Ser Asn Thr Leu Ser Ser
65 70 75 80
Pro Asn Ser Lys Asn Glu Lys Ala Leu Gly Arg Lys Ile Asn Ser Trp
85 90 95
Glu Ser Ser Arg Ser Gly His Ser Phe Leu Ser Asn Leu His Leu Arg
100 105 110
Asn Gly Glu Leu Val Ile His Glu Lys Gly Phe Tyr Tyr Ile Tyr Ser
115 120 125
Gln Thr Tyr Phe Arg Phe Gln Glu Glu Ile Lys Glu Asn Thr Lys Asn
130 135 140
Asp Lys Gln Met Val Gln Tyr Ile Tyr Lys Tyr Thr Ser Tyr Pro Asp
145 150 155 160
Pro Ile Leu Leu Met Lys Ser Ala Arg Asn Ser Cys Trp Ser Lys Asp
165 170 175
Ala Glu Tyr Gly Leu Tyr Ser Ile Tyr Gln Gly Gly Ile Phe Glu Leu
180 185 190
Lys Glu Asn Asp Arg Ile Phe Val Ser Val Thr Asn Glu His Leu Ile
195 200 205
Asp Met Asp His Glu Ala Ser Phe Phe Gly Ala Phe Leu Val Gly
210 215 220
<210> 8
<211> 178
<212> PRT
<213> Homo sapiens
<400> 8
Lys Glu Lys Gln Gln Asn Ile Ser Pro Leu Val Arg Glu Arg Gly Pro
1 5 10 15
Gln Arg Val Ala Ala His Ile Thr Gly Thr Arg Gly Arg Ser Asn Thr
20 25 30
Leu Ser Ser Pro Asn Ser Lys Asn Glu Lys Ala Leu Gly Arg Lys Ile
35 40 45
Asn Ser Trp Glu Ser Ser Arg Ser Gly His Ser Phe Leu Ser Asn Leu
50 55 60
His Leu Arg Asn Gly Glu Leu Val Ile His Glu Lys Gly Phe Tyr Tyr
65 70 75 80
Ile Tyr Ser Gln Thr Tyr Phe Arg Phe Gln Glu Glu Ile Lys Glu Asn
85 90 95
Thr Lys Asn Asp Lys Gln Met Val Gln Tyr Ile Tyr Lys Tyr Thr Ser
100 105 110
Tyr Pro Asp Pro Ile Leu Leu Met Lys Ser Ala Arg Asn Ser Cys Trp
115 120 125
Ser Lys Asp Ala Glu Tyr Gly Leu Tyr Ser Ile Tyr Gln Gly Gly Ile
130 135 140
Phe Glu Leu Lys Glu Asn Asp Arg Ile Phe Val Ser Val Thr Asn Glu
145 150 155 160
His Leu Ile Asp Met Asp His Glu Ala Ser Phe Phe Gly Ala Phe Leu
165 170 175
Val Gly
<210> 9
<211> 2421
<212> DNA
<213> Homo sapiens
<400> 9
caccctcatg agccccgggt acgtttaact attgagggcc aggaaattgc cttcctcctg 60
gacactggcg cagccttctc agggttaatc tcctgtcctg gatgactgtc ttcaaggtcc 120
gttaccaccc gaggaatcct gggacagcct ataaccaggt atttctccca catcctcagt 180
tgtaattgag agactttaat cttttcacat gccttttttg ttattcctga aagtcccaca 240
cccttattaa ggagggatat attagccaag gctggagcta ttatctacat gaatatgggg 300
aaaaagttac ccatttgctg tcccctactt gaggagggaa tcaaccctga agtctgggca 360
ttggaaggac aatttggaag ggcaaaaaat gcctgcccag tccaaatcag gttaaaagat 420
cccaccactt ttccgtatca aaggcaatat cccttaaggc ctgaagctca taaaggatta 480
tagaatattg ttaaacattt aaaagctcaa ggcttagtga ggaaatgcag cagtccctgc 540
aacaccccag ttctaggagt acaaaaacca aacagtcagt ggagactagt gcaagatctt 600
agactcatta atgaggcagt aattcctcta tatccagttg tacccaaccc ctataccctg 660
ctctctcaaa taccagggga agcagaatgg ttcacggttc tggacctcaa ggatgccttc 720
ttctttattc ccctgcactc tgactcccag tttctctttg cttttgagga tcccacagac 780
cacacgtccc aacttacaca gatggtcttg ccccaagggt ttagggatag ccctcatctg 840
tttggtcagg cactggccca agatctatag gccacttctc aagtccaggc actctggtcc 900
ttcaatatgt ggatgattta cttttggcta ccagtttgga agcctcgtgc cagcaggcta 960
ctctggatct cttgaacttt ctggctaatc aagggtacaa ggtgtctagg tcgaaggccc 1020
agctttgcct acagcaggtt aaatatctaa gcctaatctt agccaaaggg accagggccc 1080
tcagcaagga atgaatacag cctatactgg cttatcctca ccctaagaca ttaaaacagt 1140
tgcggggatt ccttggaatt actggctttt gctgactatg gatctccaga tacagcgaga 1200
cagccaggcc cctctatact ctaatcaagg aaacccagag ggcaaatact catctagtcg 1260
aaagggaacc agaggcagaa acagccttca aaagcttaaa gcaggctcta gtacaagctc 1320
cagctttaag ccttcccaca ggacagaact tctctttata catcacagag agagccaaga 1380
tagctcttgg agtccttaga ctcgtgggac aaccccacaa ccagtggcat acctaagtaa 1440
ggaaattgat gtagtagcaa aaggctggcc tcactgttta agggtagttg cagcagcggc 1500
cgtcttagcg tcagaggcta tcagaataat acaaggaaag gatctcactg tctggactac 1560
tcatgatgta aatggcatac taggtgccaa aggaagttta tggctatcag acaaccgcct 1620
cttagatgcc aggcactact ccttgaggga ctggtgctta aaatatgcac gtgcgtggcc 1680
ctcaaccctg ccacttttct cccagaggat ggggaaccaa ttgagcatga ctgccaacaa 1740
attatagtcc agacttatgc cgcccgagat gatctcttag aagtcccctt aactaatcct 1800
gaccttaacc tatataccga cggaagttca tttgtggaga atgggatacg aagggcaggt 1860
tacgccatag tgatgtaacc acacttgaaa gcaagcctct tcccccaggg accagtgccc 1920
agttagcaga actagtggca cttacccgag ccttagaact gggaaaggga aaaagaataa 1980
atgtgtatac agataacaag tatgcttatc taatcctaca tgcccatgct gcaatatgga 2040
aagaatggga gttcctaacc tctgggaacc cccgctggat gccacaggga agttatggag 2100
ttattgcaca tggtgcagga acccaaagag gtgggagtct tacactacca aggccatcaa 2160
aatgggaagg agaggggaga acagcagcat aagcggctgg cagaggtagg gaaagaccag 2220
caagaaggaa agagagaaag agaaagtcag agaaagagac agagagagga agagacagag 2280
agacagaacg ttaaagaggg tgtcagaaac agagacaaac aaaaggagtc agaaagaagg 2340
acagacacag aaagtcaaag agagagttaa aaagagagga agagacaaag aagtcgaaga 2400
gagaaagaga gagatggaag t 2421
<210> 10
<211> 2421
<212> DNA
<213> Homo sapiens
<400> 10
ctttgcagat aaatatggca cactagcccc acgttttctg agacattcct caattgctta 60
gacatattct gagcctacag cagaggaacc tccagtctca gcaccatgaa tcaaactgcc 120
attctgattt gctgccttat ctttctgact ctaagtggca ttcaaggtaa ggaacatcaa 180
aggatactta atttgtaaaa tgagaaatag gaataggtat aaattctaaa aatacagaaa 240
taatgtattt gtaaaagttt cactgcatgc ttataaataa gagggaaata aatagagatt 300
ccctcagatc ataaaactta tatgaattga agtgagagaa acaaatagaa taagagaaag 360
agaaggaaaa agggaaggag gacagaagag atggggaaga gggaggatag agagagaaaa 420
tgtgagggaa tgcggacaga gatgagatac agatacttcc ttacctaact aagctcaatg 480
aaccacatga actgtgctta agggtttgac tttataatca acaagctgca attcttttct 540
tccagataat caactcttta atcatttaca gttgtgttat gatgtgatcc attcctcctc 600
agattaagtg actatttgct gatatgggga tataggttct gctaaatacc accagtctac 660
attaaatgcc taaaatgaac actgtgctaa ccttctctgc tgttcctctt ttcctacagg 720
agtacctctc tctagaactg tacgctgtac ctgcatcagc attagtaatc aacctgttaa 780
tccaaggtct ttagaaaaac ttgaaattat tcctgcaagc caattttgtc cacgtgttga 840
gatcatgtga gtgaaatccc atctgattat cacttccctg gttgtaatta tatactgtat 900
taaatatgta atgataataa aaaaagatca gtaaagggtt tgtgatgatt ctaaaactaa 960
tgtacagcaa acaaaaacat gcagagtgaa acttaaatgt ctgacttcag aactgcgtat 1020
gccatctgtt ttattgaccc aacacagttt taaatatttt catccctatt tatttctaca 1080
gtgctacaat gaaaaagaag ggtgagaaga gatgtctgaa tccagaatcg aaggccatca 1140
agaatttact gaaagcagtt agcaaggaaa ggtaggtttg ctgttgcctg cagaagaatt 1200
gctctttagg aaacggcaat cttgggagtc agaaatactt gcattgtggt ttgctgtgca 1260
atcgctggtt taaaagtatg ttaccaccac gccctcccct acctccattt atttaaatgc 1320
tgaggcacca tcttgtgtga taagtatcag aagttaccct gattaccagt caaccttgaa 1380
gtacagctat aactatctaa gcaaaactga caacattttc cccaagtctt tcatggttga 1440
aaaaagcaac ccctataatc cataatgaat gcatagcagc aggaaagctc agttatctat 1500
tctatgaact cggtactttc caaacacaac ccaatctgaa gccagagtca gactatcaca 1560
cttttatatc ccctttctct tcttacaggt ctaaaagatc tccttaaaac cagaggggag 1620
caaaatcgat gcagtgcttc caaggatgga ccacacagag gctgcctctc ccatcacttc 1680
cctacatgga gtatatgtca agccataatt gttcttagtt tgcagttaca ctaaaaggtg 1740
accaatgatg gtcaccaaat cagctgctac tactcctgta ggaaggttaa tgttcatcat 1800
cctaagctat tcagtaataa ctctaccctg gcactataat gtaagctcta ctgaggtgct 1860
atgttcttag tggatgttct gaccctgctt caaatatttc cctcaccttt cccatcttcc 1920
aagggtacta aggaatcttt ctgctttggg gtttatcaga attctcagaa tctcaaataa 1980
ctaaaaggta tgcaatcaaa tctgcttttt aaagaatgct ctttacttca tggacttcca 2040
ctgccatcct cccaaggggc ccaaattctt tcagtggcta cctacataca attccaaaca 2100
catacaggaa ggtagaaata tctgaaaatg tatgtgtaag tattcttatt taatgaaaga 2160
ctgtacaaag tagaagtctt agatgtatat atttcctata ttgttttcag tgtacatgga 2220
ataacatgta attaagtact atgtatcaat gagtaacagg aaaattttaa aaatacagat 2280
agatatatgc tctgcatgtt acataagata aatgtgctga atggttttca aaataaaaat 2340
gaggtactct cctggaaata ttaagaaaga ctatctaaat gttgaaagat caaaaggtta 2400
ataaagtaat tataactaag a 2421
<210> 11
<211> 2421
<212> DNA
<213> Homo sapiens
<400> 11
agatgacttt tttctattta tatttaataa gatgatgaac ccttcttgca ttcccgaaat 60
aaacctcaac tgttacagtg ttttattctt ttaatatgta cgaagtacat gttaagcaag 120
ttatttccta agcagcccca caaactgggc actactacca tcctgctctg cccctccctc 180
actctacttc agccacttca gccacaatgg cctctcctca ctgcccctct gatgcaccaa 240
gcttgttctc acctcaggaa tgtgcaacac ctgccagact tgctgttccc cggagcctcc 300
atccccagat atcctcatat atcatcctcc tcttcatttg tgtctctgct tacatatgac 360
ctatttacag aagcctttcc tgtctacccc ccatgaaata gaaatcgcat tccaatcttg 420
tctctacccc aatgctgttt cattttgtct gtagcaattg tcatcatctc atatatattc 480
acatgtggaa aatacacaaa atgtttaact tcttttaatt tacattccat ttccccatga 540
attgaagctc catgacagcg gagatttttc tctgctttcc ctgttgctca cttcccagca 600
ccaagagcag gcctggcaca tgggaagtac ttactattta ttgaatggat gaatgaacaa 660
atgaatgaat ggatacttat tttacacata agaaaactga agcttataga aattaagtaa 720
ctaaaatcac acagaagcac agctgaaact aaaacctacg tctaactttc aattcctgac 780
ccttaaccat taaaacaaat gacaggtgac tttaggccac tgaaaatgct catataatct 840
tatgaattct aaagcacaag ttaatcacac cattgattga aagtctgagg aatactgtat 900
agacaagccc ctgtacaagg taagcaaaag aatcagagga tggcctccaa agaattccct 960
ggacattatg ggaattacat tgttagcctt cctactgata cccataagcc tcacagcaag 1020
catcatgaag ctgtgacctt catctgcaca tgcccttgta tacccaaaag ataaaactgg 1080
atgcttcagg gccgaatggc caataaacac gtgtttatta ctggcatggg cagacacaca 1140
tactgaaagt accatttccc agcggactag ccatattatg atcagtacag acactaaaga 1200
tttagctttg aaaaaactat ttgctcttcc aaagctgaag aatcttctgt gatttcaaca 1260
ggcaagttac agtcaggtat tcttaatgtt cttttcctcc tctctcactg ggatactttc 1320
tttccttcag acaacgtcaa gcgaaaaaca aaatttcaca aatctccatt tctgacacta 1380
aacagtacag tatctttatt ttttttataa tttaatcaaa ccctgtattt tagaactgtg 1440
gggctgatcc aacattgcaa tgtgtcacat ttaattccat caatgtaaag cataatgagc 1500
aaagattaag gtagtgaggc ataactaaat gttttgaacc tgtgaatttc aaaagcaagg 1560
cccatttgtg ttattttcta aatagtaaat aaaatcattt tccaacattt cactatcaaa 1620
ttacagtaat ttttccacca gtacacactt gaggaaagcc acaaaaagac ttttccaaca 1680
gttcattctg ttattgctca taaccttcta aatacttctc ctcattggct tctattcaaa 1740
ggtaaatgga aagcagtaaa atttatggaa aatatattca actgcttaaa atacatcaac 1800
caaaaaaaag attttagagc tgtattatga gttgtgaaat tgcattgcct tcacttacct 1860
ttcagtttca ctggtaggta acaaaactga cagactggtc aagttccaaa acatccccta 1920
tatagagcct gactcttcca tctcaaattc tcaccttggt caaggccaga gtaaacacct 1980
gtccttcaca tttttacaca acatcacttt gtatgctaca aatataagct ttcataccag 2040
ggaggaagca aattccagga cactggaaac atttctgctc tcttaaacca gtctgttgat 2100
tgttcccttg actttctcag ctgtcaggat agtgaaagga ggaaaactgc aaaactgtaa 2160
agtataacct gataagtttg ccctttaagc ttttcacaca gagagaggta aaataaaact 2220
caagtctaag gtttaaaatt gagctatgaa tattatattc tagcactaga caaaaatgtt 2280
gcaagatttt aataaaataa gattattaaa atcaattttt acatttcatg ggccaaggag 2340
agacatcaaa gaatgtttaa ctaacatttt aaagatacta tactttataa agttaagaag 2400
aaaaatgaca actgcaccag t 2421
<210> 12
<211> 1227
<212> DNA
<213> Homo sapiens
<400> 12
ctttgcagat aaatatggca cactagcccc acgttttctg agacattcct caattgctta 60
gacatattct gagcctacag cagaggaacc tccagtctca gcaccatgaa tcaaactgcc 120
attctgattt gctgccttat ctttctgact ctaagtggca ttcaaggagt acctctctct 180
agaactgtac gctgtacctg catcagcatt agtaatcaac ctgttaatcc aaggtcttta 240
gaaaaacttg aaattattcc tgcaagccaa ttttgtccac gtgttgagat cattgctaca 300
atgaaaaaga agggtgagaa gagatgtctg aatccagaat cgaaggccat caagaattta 360
ctgaaagcag ttagcaagga aaggtctaaa agatctcctt aaaaccagag gggagcaaaa 420
tcgatgcagt gcttccaagg atggaccaca cagaggctgc ctctcccatc acttccctac 480
atggagtata tgtcaagcca taattgttct tagtttgcag ttacactaaa aggtgaccaa 540
tgatggtcac caaatcagct gctactactc ctgtaggaag gttaatgttc atcatcctaa 600
gctattcagt aataactcta ccctggcact ataatgtaag ctctactgag gtgctatgtt 660
cttagtggat gttctgaccc tgcttcaaat atttccctca cctttcccat cttccaaggg 720
tactaaggaa tctttctgct ttggggttta tcagaattct cagaatctca aataactaaa 780
aggtatgcaa tcaaatctgc tttttaaaga atgctcttta cttcatggac ttccactgcc 840
atcctcccaa ggggcccaaa ttctttcagt ggctacctac atacaattcc aaacacatac 900
aggaaggtag aaatatctga aaatgtatgt gtaagtattc ttatttaatg aaagactgta 960
caaagtagaa gtcttagatg tatatatttc ctatattgtt ttcagtgtac atggaataac 1020
atgtaattaa gtactatgta tcaatgagta acaggaaaat tttaaaaata cagatagata 1080
tatgctctgc atgttacata agataaatgt gctgaatggt tttcaaaata aaaatgaggt 1140
actctcctgg aaatattaag aaagactatc taaatgttga aagatcaaaa ggttaataaa 1200
gtaattataa ctaagaaaaa aaaaaaa 1227
<210> 13
<211> 98
<212> PRT
<213> Homo sapiens
<400> 13
Met Asn Gln Thr Ala Ile Leu Ile Cys Cys Leu Ile Phe Leu Thr Leu
1 5 10 15
Ser Gly Ile Gln Gly Val Pro Leu Ser Arg Thr Val Arg Cys Thr Cys
20 25 30
Ile Ser Ile Ser Asn Gln Pro Val Asn Pro Arg Ser Leu Glu Lys Leu
35 40 45
Glu Ile Ile Pro Ala Ser Gln Phe Cys Pro Arg Val Glu Ile Ile Ala
50 55 60
Thr Met Lys Lys Lys Gly Glu Lys Arg Cys Leu Asn Pro Glu Ser Lys
65 70 75 80
Ala Ile Lys Asn Leu Leu Lys Ala Val Ser Lys Glu Arg Ser Lys Arg
85 90 95
Ser Pro
<210> 14
<211> 2301
<212> DNA
<213> Homo sapiens
<400> 14
aaggcaagag atctaggact tctagcccct gaactttcag ccgaatacat cttttccaaa 60
ggagtgaatt caggcccttg tatcactggc agcaggacgt gaccatggag aagctgttgt 120
gtttcttggt cttgaccagc ctctctcatg cttttggcca gacaggtaag ggccacccca 180
ggctatggga gagatttgat ctgaggtatg ggggtggggt ctaagactgc atgaacagtc 240
tcaaaaaaaa aaaaaaaaga ctgtatgaac agaacagtgg agcatccttc atggtgtgtg 300
tgtgtgtgtg tgtgtgtgtg tgtgtgtgtg tggtgtgtaa ctggagaagg ggtcagtctg 360
tttctcaatc ttaaattcta tacgtaagtg aggggataga tctgtgtgat ctgagaaacc 420
tctcacattt gcttgttttt ctggctcaca gacatgtcga ggaaggcttt tgtgtttccc 480
aaagagtcgg atacttccta tgtatccctc aaagcaccgt taacgaagcc tctcaaagcc 540
ttcactgtgt gcctccactt ctacacggaa ctgtcctcga cccgtgggta cagtattttc 600
tcgtatgcca ccaagagaca agacaatgag attctcatat tttggtctaa ggatatagga 660
tacagtttta cagtgggtgg gtctgaaata ttattcgagg ttcctgaagt cacagtagct 720
ccagtacaca tttgtacaag ctgggagtcc gcctcaggga tcgtggagtt ctgggtagat 780
gggaagccca gggtgaggaa gagtctgaag aagggataca ctgtgggggc agaagcaagc 840
atcatcttgg ggcaggagca ggattccttc ggtgggaact ttgaaggaag ccagtccctg 900
gtgggagaca ttggaaatgt gaacatgtgg gactttgtgc tgtcaccaga tgagattaac 960
accatctatc ttggcgggcc cttcagtcct aatgtcctga actggcgggc actgaagtat 1020
gaagtgcaag gcgaagtgtt caccaaaccc cagctgtggc cctgaggccc agctgtgggt 1080
cctgaaggta cctcccggtt ttttacaccg catgggcccc acgtctctgt ctctggtacc 1140
tcccgctttt ttacactgca tggttcccac gtctctgtct ctgggccttt gttcccctat 1200
atgcattgca ggcctgctcc accctcctca gcgcctgaga atggaggtaa agtgtctggt 1260
ctgggagctc gttaactatg ctgggaaacg gtccaaaaga atcagaattt gaggtgtttt 1320
gttttcattt ttatttcaag ttggacagat cttggagata atttcttacc tcacatagat 1380
gagaaaacta acacccagaa aggagaaatg atgttataaa aaactcataa ggcaagagct 1440
gagaaggaag cgctgatctt ctatttaatt ccccacccat gacccccaga aagcaggagg 1500
gcattgccca cattcacagg gctcttcagt ctcagaatca ggacactggc caggtgtctg 1560
gtttgggtcc agagtgctca tcatcatgtc atagaactgc tgggcccagg tctcctgaaa 1620
tgggaagccc agcaatacca cgcagtccct ccactttctc aaagcacact ggaaaggcca 1680
ttagaattgc cccagcagag cagatctgct ttttttccag agcaaaatga agcactaggt 1740
ataaatatgt tgttactgcc aagaacttaa atgactggtt tttgtttgct tgcagtgctt 1800
tcttaatttt atggctcttc tgggaaactc ctcccctttt ccacacgaac cttgtggggc 1860
tgtgaattct ttcttcatcc ccgcattccc aatataccca ggccacaaga gtggacgtga 1920
accacagggt gtcctgtcag aggagcccat ctcccatctc cccagctccc tatctggagg 1980
atagttggat agttacgtgt tcctagcagg accaactaca gtcttcccaa ggattgagtt 2040
atggactttg ggagtgagac atcttcttgc tgctggattt ccaagctgag aggacgtgaa 2100
cctgggacca ccagtagcca tcttgtttgc cacatggaga gagactgtga ggacagaagc 2160
caaactggaa gtggaggagc caagggattg acaaacaaca gagccttgac cacgtggagt 2220
ctctgaatca gccttgtctg gaaccagatc tacacctgga ctgcccaggt ctataagcca 2280
ataaagcccc tgtttacttg a 2301
<210> 15
<211> 2301
<212> DNA
<213> Homo sapiens
<400> 15
aggaattgaa ctcagctctg ccccaagcgg acctaataga catctacaga actctccacc 60
ccaaatcaac agaatataca tttttttcag caccacacca cacctattcc aaaattgatc 120
acatagttgg cagtaaagct ctcctcagca aatgtaaagg aacagaaatt ataacaaact 180
atctctcaga ccacagtgca atcaaattag aactcagaat taagaatctc actcaaaacc 240
gcacaactac atggaaactg aacaacctgc ttctgaatga ctactgggta cataatgaaa 300
tgaaggcaga aataaagatg ttctttgaaa tgaacaagaa caaacacaca acataccaga 360
atctctggga cgcattcaaa gcagtgtgta gagggaaatt tatagcacta aatgcccaca 420
agagaaagca ggaaacatcc aaaattgaca tcctaacatc acagttaaaa gaactagaaa 480
agcaagagca aacacattca aaagctagca gaaggcaaga gataactaaa atcagagcag 540
aactgaagga aatagagaca caaaaaccct tcaaaaaatt aatgaatcca ggagctggtt 600
ttttgaaagg atcaacaaaa tagatagacc actagcaaga ctaataaaga aaaaaagaga 660
gaagaatcaa atagacacaa taaaaaaatg ataaagggga tatcaccacc gatcccacgg 720
aaatacaaac taccatcaga gaatactaca aacacctcta cgcaaataaa ctagaaaatc 780
aagaagaaat ggataaattc ctcgacacat acactctccc aagactaaac caggaagaag 840
ttgaatctct gaatagacca ataacaggat atgaaattgt ggcaataatc aataccttac 900
caacaaaaaa gagtccagga ccagatggat tcacagccga attctaccag aggtacaagg 960
aggaactggt accattcctt ctgaaactat tccaatcaat agaaaaagag ggaatcctcc 1020
ctaactcatt ttatgaggcc agcatcattc tgataccaaa gccgggcaga gacacaacca 1080
aaaaagagaa ttttagacca atcaatatcc ttgatgaaca ttgatgcaaa aatcctcaat 1140
aaaatactgc caaaccaaat ccagcagcac atcaaaaagc ttatccacca tgatcaagtg 1200
ggcttcatcc ctgggatgca aggctggttc aatatacgca aatcaataaa tgtaatccag 1260
catataaaca gagccaaaga caaaaaccac atgattatct caatagatgc agaaaagacc 1320
tttgacaaaa ttcaacaacc cttcatgctc aaaactctca ataaattagg tattgatggg 1380
acgtatttca aaataataag agctatctat gacaaaccca cagccaatat catactgaat 1440
gggcaaaaac tggaagtatt cactttgaaa actggcacaa gacagggatg ccctctctca 1500
ccactcctat tcaacatagt gttggaagtt ctggccaggg caattaggca ggagaaggaa 1560
ataaagggta ttcaattagg aaaagaggaa gtcaaattgt ccctgtttgc agacgacatg 1620
attgtatatc tagaaaaccc cattgtctca gcccaaaatc tccttaagca gataagcaac 1680
ttcagcaaaa tctcaggata caaaatcaat gtacaaaaat cacaagcatt cttatacacc 1740
aacaacagac aaacagagag ccaaatcatg agtgaaatcc cattcacaat tgctttaaag 1800
agaataaaat acctaggaat ccaacttaca agggatgtga aggacctctt caaggagaac 1860
tacaaaccac tgctcaatga aataaaagag gataaaaaca aatggaagaa cattccatgc 1920
tcatgggtag gaagaatcaa tatcatgaaa atggccatac tgcccaaggt aatttacaga 1980
ttcaatgcca tccccatcaa gctaccaatg cctttcttca cagaattgga aaaaactatt 2040
tttagttcat atggaaccaa aaaagagccc gcattgccaa gtcaatccta agccaaaaga 2100
acaaagctgg aggcatcaca ctacctgact tcaaactata ctacaaggct acagtaacca 2160
aaacagcatg gtactggaac caaaacagag atatagatca atggaacaga acagagccct 2220
caaaattaat gccacatatc tacaactatc tgatctttga caaacctgag aaaaaccagc 2280
aatggggaaa ggattcccca t 2301
<210> 16
<211> 2024
<212> DNA
<213> Homo sapiens
<400> 16
aaggcaagag atctaggact tctagcccct gaactttcag ccgaatacat cttttccaaa 60
ggagtgaatt caggcccttg tatcactggc agcaggacgt gaccatggag aagctgttgt 120
gtttcttggt cttgaccagc ctctctcatg cttttggcca gacagacatg tcgaggaagg 180
cttttgtgtt tcccaaagag tcggatactt cctatgtatc cctcaaagca ccgttaacga 240
agcctctcaa agccttcact gtgtgcctcc acttctacac ggaactgtcc tcgacccgtg 300
ggtacagtat tttctcgtat gccaccaaga gacaagacaa tgagattctc atattttggt 360
ctaaggatat aggatacagt tttacagtgg gtgggtctga aatattattc gaggttcctg 420
aagtcacagt agctccagta cacatttgta caagctggga gtccgcctca gggatcgtgg 480
agttctgggt agatgggaag cccagggtga ggaagagtct gaagaaggga tacactgtgg 540
gggcagaagc aagcatcatc ttggggcagg agcaggattc cttcggtggg aactttgaag 600
gaagccagtc cctggtggga gacattggaa atgtgaacat gtgggacttt gtgctgtcac 660
cagatgagat taacaccatc tatcttggcg ggcccttcag tcctaatgtc ctgaactggc 720
gggcactgaa gtatgaagtg caaggcgaag tgttcaccaa accccagctg tggccctgag 780
gcccagctgt gggtcctgaa ggtacctccc ggttttttac accgcatggg ccccacgtct 840
ctgtctctgg tacctcccgc ttttttacac tgcatggttc ccacgtctct gtctctgggc 900
ctttgttccc ctatatgcat tgcaggcctg ctccaccctc ctcagcgcct gagaatggag 960
gtaaagtgtc tggtctggga gctcgttaac tatgctggga aacggtccaa aagaatcaga 1020
atttgaggtg ttttgttttc atttttattt caagttggac agatcttgga gataatttct 1080
tacctcacat agatgagaaa actaacaccc agaaaggaga aatgatgtta taaaaaactc 1140
ataaggcaag agctgagaag gaagcgctga tcttctattt aattccccac ccatgacccc 1200
cagaaagcag gagggcattg cccacattca cagggctctt cagtctcaga atcaggacac 1260
tggccaggtg tctggtttgg gtccagagtg ctcatcatca tgtcatagaa ctgctgggcc 1320
caggtctcct gaaatgggaa gcccagcaat accacgcagt ccctccactt tctcaaagca 1380
cactggaaag gccattagaa ttgccccagc agagcagatc tgcttttttt ccagagcaaa 1440
atgaagcact aggtataaat atgttgttac tgccaagaac ttaaatgact ggtttttgtt 1500
tgcttgcagt gctttcttaa ttttatggct cttctgggaa actcctcccc ttttccacac 1560
gaaccttgtg gggctgtgaa ttctttcttc atccccgcat tcccaatata cccaggccac 1620
aagagtggac gtgaaccaca gggtgtcctg tcagaggagc ccatctccca tctccccagc 1680
tccctatctg gaggatagtt ggatagttac gtgttcctag caggaccaac tacagtcttc 1740
ccaaggattg agttatggac tttgggagtg agacatcttc ttgctgctgg atttccaagc 1800
tgagaggacg tgaacctggg accaccagta gccatcttgt ttgccacatg gagagagact 1860
gtgaggacag aagccaaact ggaagtggag gagccaaggg attgacaaac aacagagcct 1920
tgaccacgtg gagtctctga atcagccttg tctggaacca gatctacacc tggactgccc 1980
aggtctataa gccaataaag cccctgttta cttgaaaaaa aaaa 2024
<210> 17
<211> 224
<212> PRT
<213> Homo sapiens
<400> 17
Met Glu Lys Leu Leu Cys Phe Leu Val Leu Thr Ser Leu Ser His Ala
1 5 10 15
Phe Gly Gln Thr Asp Met Ser Arg Lys Ala Phe Val Phe Pro Lys Glu
20 25 30
Ser Asp Thr Ser Tyr Val Ser Leu Lys Ala Pro Leu Thr Lys Pro Leu
35 40 45
Lys Ala Phe Thr Val Cys Leu His Phe Tyr Thr Glu Leu Ser Ser Thr
50 55 60
Arg Gly Tyr Ser Ile Phe Ser Tyr Ala Thr Lys Arg Gln Asp Asn Glu
65 70 75 80
Ile Leu Ile Phe Trp Ser Lys Asp Ile Gly Tyr Ser Phe Thr Val Gly
85 90 95
Gly Ser Glu Ile Leu Phe Glu Val Pro Glu Val Thr Val Ala Pro Val
100 105 110
His Ile Cys Thr Ser Trp Glu Ser Ala Ser Gly Ile Val Glu Phe Trp
115 120 125
Val Asp Gly Lys Pro Arg Val Arg Lys Ser Leu Lys Lys Gly Tyr Thr
130 135 140
Val Gly Ala Glu Ala Ser Ile Ile Leu Gly Gln Glu Gln Asp Ser Phe
145 150 155 160
Gly Gly Asn Phe Glu Gly Ser Gln Ser Leu Val Gly Asp Ile Gly Asn
165 170 175
Val Asn Met Trp Asp Phe Val Leu Ser Pro Asp Glu Ile Asn Thr Ile
180 185 190
Tyr Leu Gly Gly Pro Phe Ser Pro Asn Val Leu Asn Trp Arg Ala Leu
195 200 205
Lys Tyr Glu Val Gln Gly Glu Val Phe Thr Lys Pro Gln Leu Trp Pro
210 215 220
<210> 18
<211> 26803
<212> DNA
<213> Homo sapiens
<400> 18
cttgctctgt cacccaggct ggagtgcagt gctgtgatca tggttcactg cagccttgaa 60
ctcctgggct ctggcaatcc tcctgcctga gccttctgag tagctgagac tatagatatg 120
ggccaccaca cctggctaat ttttaatttt ttttagtaga gatgaagtct tgctatgttg 180
accaggcttg tgggagttca gtcaggctgg tggaaaaaat tttaaagata gttataagaa 240
atagacacaa accttcttgt aaggctggag agggtttaca ttgcttcagt aacagatttg 300
gctgaaagca gcctaatcct ctctaccttt agctgatagc aaaaatgaaa ataacaaggg 360
aatgtgagga agtttatcta aatagcttgc ttactcatgt ggtcctaaaa ccaaactttg 420
atcaacctca ggtgcataat tgctctctac tcagggggtg agcaatgtta attaccctct 480
agtggtgttt actcgagacc tttgtcattt aatctgtatt aaataaatgt gaactttgct 540
agcttattga ggtgatgctc cagatgcaga gcagagcccc ttagccagac tgacaggcaa 600
aatatctgtg tcagtgtatg tctctcatcc atcactggtt cagggtctgc gggctggatt 660
cctgcacagg ctggtcttga actcctgggc tcaagcaatc ctcccgcctg agccttctga 720
gtagctgaga ctacagatat gggccaccac acctggctat ttttaacatt ttttagtaga 780
gatgaagtct tgctgtgttg cccaggctgg tcttaaactc ctgggctcaa gagctcctcc 840
tgccttggcc tcccaaagtg ctgggagtac aggcatgagt catggtgccc agacggacat 900
ttttttaaaa ataaaggaat actcctgaaa cgctgaagtc ttctttgtac ccctctgtga 960
taacatgaat agcctcttaa tgacaccaag gagcaagaca agttagtccc aaagtagctc 1020
acatagatga tgataaagga atgaggggtg ggtgtgatct atgcaaaaaa cccttactct 1080
ttaatgggtt gctgtttcta acataattgc aacagtaccc tacattgcta taatgcatta 1140
tagttttcaa agtgcttttg tggcctccat aatagagagg ttgtgaggta agcttcaacc 1200
acggctgccc tactacccaa gagtggacac acaatgatga ataggtcaat ttctgcctct 1260
tagtttctta gctagggagg ggcacttact ggaacagcac agaaaacaga gtctttggcc 1320
cagttggaga atctttatca ggttatgcta cttcaagctt tcctcctgtt aaatgtgaga 1380
ggaataaatc cctgttcctt ggatcagtgt gactctgaaa cagctggagg agagacagct 1440
ttaggcaggt tgaaacagag agctccagca tatgtacttt tttttttttt ttgagatgga 1500
gtctcgctct gtctcccagg ctggagtgca gtggctcgat cttggctcac tgcaagctct 1560
gcctcccagg ttcatgccat tctcccgcct cagcctccct agtagctggg actacaggct 1620
cccgccacca cgcccggcta atttttttgt atttttagta gagacagggt ttcaccatgt 1680
tggccaggcc agtcttgaac tcctgacctg aggtgatcca cctgcctcag tctcccaaag 1740
tgctgggatt acaggcgtga gccactgaac ttggccaagt gcactacttt taaaagttaa 1800
agtattatgc agccatgagg gaatattgtg caagaagaaa gcttttacaa gaaaaacttg 1860
aaacattggt atttttgcct cctttttaac aactagagtc gttttgggag ttgtttcttg 1920
tcgaagaaac aacccatgtt tattttccca gtatggcagg accatgtagg aaagcaaaat 1980
tacccctcag gagaggaaat tctctgacac tctataaggc tccataaccc tcctctgaac 2040
tgtggccaac aagattgggt agcacttttt aaggtagttt aagaaaaata ggctgtgcat 2100
ggtggtttat gcctgtaatc ccagcacttt gggaggccga ggtgagtgga tcacctgagg 2160
tcaggagttt gagaccagcc tgaccaatat ggtgaaaccc catctctact gaaaatacaa 2220
aaattagccg ggtgtggtgg cgggcacctg tagtcccagc tactcgggag gctgaggcag 2280
gagaattgct tgaacctggg aggcggaggt tgcagtgagc cgagactgtg ccactgcact 2340
ccagcctggg tgacagagca ggactccatc tcaaaaaaaa aaaaaaaaaa aagaaagaaa 2400
gaaaaatata tagtgagccc aataaagctg tataatctaa atcaaacatg acttgcatgc 2460
ctggagactt tgcactagaa aaatatttct acctaaaaaa tcaaatttta ttttctttct 2520
cacaaatatt caatctgctt tcatctcagt tcttcctacc ttgtcaaacc tctccccaca 2580
tttcctattc tttttctctc cagcctgata tctctcatta tactgcttaa gagaaatgtt 2640
atgttactat tcttttctcc cagaactgtt tctctggttc tttaaggtgt ctgagtacac 2700
actgtgcctt cttcctttta gccctctctt ctccttgttc cctgagcctt acctttatga 2760
ccttagaact tcaagttccc actacaattt taaatataga ctattttgct tttcctccta 2820
ctagggagct taaattgcct ctaattacac tgttttcccg agtccttcct ctcctttgca 2880
atttagatat agcacagaag cacattttgc ttgactgtcc tctgaactgt catgctgttt 2940
tgatgtggtt ctattgtcca agagtcttgg ttaaataata gcccagcatc ccacctgtgt 3000
ttaaaagaac tgcttcacag gcaaatcaaa aagcccatgg atccgaagtc acaatgggcg 3060
ttgctattca aacagcacca gattgctatt caacgcttgg ttgaaaaata aatttcagtt 3120
tcattcacct aatatatttc cttctatttt gtaggatggt atgctcttac ttcaatttgg 3180
acttgttcac aattgaacgt taacatagcc ttatagatta gcctgttttc attggtccag 3240
agtattctcc aaataaagca gggtctgtgc attttaagca actcccctga acaatttcat 3300
gggcattctc aaatttgaga actacctgaa aagctgtgtt ggagaaaaga acaaccaatg 3360
aatgtggcag gacagaatat tgaacattaa cttccctttt cctcttctcc catctgttct 3420
cttccattat ccctacccgt ccgctcagtc tcgttattca ggcaacagtt attttgccat 3480
tatttcctca agaaaggaac aaaagtaaac acaattgctt tctgattttt tttttttttt 3540
tgcattttaa aatggacttt gaaaccataa gcaaagaggt gtttaagagt ctttccaaag 3600
ccaaaaatga aggttttgaa atttcaaagt cactgccttg aagagactcg aggtttggag 3660
tgtgtacagt atgtcggagc tggacttttc tccttcctga gactagataa cggtctgaat 3720
ccaagacagt tttcatgatt tcagaggaag tggtcaagtg gtctgtgagg tagaccttct 3780
gcttaagagc agtcaggagg ccgggtgccg tggctcacgc ctgtaatccc agcactttgg 3840
gagaccgagg tgggcggatc acctgaggtc aggagttcaa gacaagcctg accaacatgc 3900
agaaaccctg tctctactaa aaatacaaaa aattggccgg gcacggtggc acatgcactc 3960
cagcctgggc aacaatagtg aaactccatc tcaaaaaaaa aacaaaaaaa agagaagaaa 4020
agaaaagaaa aggagcagtc aggatgtgtg cctccaaagc tgaggtagac aaaaagatac 4080
cagagttcta gaggcctgcc aggcacagca gcagcagcag aaggaaggtg tgggcgagaa 4140
cagggcagcc aggcgtgtgc cacctcccag acacaattat tgggaatgga gggcaagtgg 4200
tgatgggaga aaatcttgac ttaattgatg tcaagattaa agaaatgcca cctggtggca 4260
tttaagttca cacataggta aagaaagtta tgcatttact gtgaaagtca tcccactatt 4320
tagtagaaac aggagatctg gattctggtc aagagtctct tttgccaact gtggcaccac 4380
tgagcagcgg cacagctttt gtgaatcctg ggttcttcat tattaaaatg gggacattag 4440
cgttgggttg agtataagaa atggacattt ttgcaggtca aaaatggttg aatatttgca 4500
ttttcatatg attcaaccga atacttactt cacaggcata aggaaaaaaa tagaataaca 4560
tactaacaac tgtccctgga gtaagtactt aacaaataca tgatttataa agaagatatg 4620
tgaaagatat ttgtaagtac atgatttata gaaagatatg aaagtatgta aacccttgtg 4680
gtctaatggt cacagaataa tctgagctta atatccctgc tccctaccat acagaaggca 4740
aaatgcctat taggggtttt ctttcttcac cctctccttc tttttcctcc tcctcttgac 4800
tcctcctcat cctcctcttt cttcttcccc ttattaatgt ctaaaaaggg gctgagcatg 4860
gtggctcatg cctgtaatcc tagcactctg ggaagctgcg gcaggtggat cacctgaggc 4920
caggagtttg aaaccagcct ggtcaacatg gcaaaacccc atccctacta aaaatacaaa 4980
aattacccag gtgtggtggc aggcacctgt aattccaact acccgggagg ctgaggcagg 5040
agaatcgctt aaacccggga ggcagaggtt gcagtgagcc aagattgtgc cactgttgtc 5100
catcctgggt gacagaggga gactctgtct caagataaat aataataata ataataattt 5160
ctaaaaaggt aatacatttt catagttcaa aaaccaaaag gtataaaagg aaatacagta 5220
aaaaatttcc tatcatatca ctgtctagag tactattcct tatatatttt cctgattttt 5280
gagtatttta aaatgtgagt gttggatatg agtgttggat ttaaaaagtt ttatgataat 5340
ttgtgtatat ttgtgtgtgt gtgtgtgtgt gtgtgtgtgt atagtagtcc aagactatca 5400
gtttatgaat aataagagga gacccatgga aaaccagtcc ctttgaccaa gttcactcag 5460
ataatcagca gcagggcttg gacattaatt acagttatcc aacatccttt gaggtctcac 5520
atgacaaatt acaaatatgg agtgtaaatg taacccactt tgctaggcaa aaaaagccct 5580
gtttttttaa aaaatatata tttttggctt atgggcaaca gaagccaggg agacgtacag 5640
tcaaacctca tttctcatgg ctttcatatc tgcaaattct cctactcatt aaaagttatt 5700
tataactccc gaatcaatac ccacagcact tttgtgatca ttggcagaca tgtgcagaaa 5760
agaaaaaaaa attgagttgt tgattgcaca cattcccagc tgaatttcaa caaagcaaca 5820
ctctgccttc ccacttcagc tttcttacta tatgtgtgtc ctttttctgt ttatttagta 5880
ccatgttttt cacactttcg ttctttttgg tggtgatttt gctgtttaaa atggccaaca 5940
agtgtagtgc taagtgctgc gtagggttct taagcacaag aaggctatga tgtgccttat 6000
ggagaaaata cgtgtgttgg atcagtttca ctcaagcatg agttatggcg ctattagctg 6060
tgagttcaat gttaacaaat caacaatata tgctaaagtg tctttaaaca gaaacacaca 6120
taaaacaagg ttatatgttt ggttggcaaa aatgttataa ccagaagctt gcagaaacct 6180
aaccctgtat ttcccttaag agcaatggtt cattattcac taattcaatg tttacagcaa 6240
ctttataaac tataactacc atgaataatg agaattgact atgttttaga tcataatacc 6300
tgaagtgaag ttttccatca tggtttttag ttcttagagt ccatttaagt tttttaaaaa 6360
tatgatagct atcctatcac atcccacatt cttgagatta aaaaattaat cttttatttg 6420
gtacagaaaa ggctcctgcg tgcatttttg gaggggctgg accgctgagg aagtcatggt 6480
tggctcaagt ggtttaaatg cattgctaat ttcatagcct ctgaaccact tatgaaacaa 6540
tactgatgat atgtcagtag ctatgtttgc agctccttca ggaatgctta tatcttttac 6600
tgctggtaaa gaagaaattt atagtaatag attttttttt tttttagata gagtctcact 6660
ctgtcgccta ggctggatta cagcggcttg atcacggttt actgcagcct ggatctccca 6720
ggctctccta ctgtagcctc cccagtaact agggccccca cacaggcatc accacaccca 6780
gctcattttt ttatgtttta tagagacagg gtcttactat gttttccagg ctggtctcaa 6840
acttctgggc tcaagcaatc ctcctacttt ggcctcccaa agtgctgaaa ttacaggcat 6900
gaaccactgc acctggttta tggtcatctt aagtagagac ttatgagtgc gtatcattgt 6960
atcaccaatc tgagactcaa ttttatcttt ctgttaatag acaatgcagt ttttctgtta 7020
attacaatga aaatcaaggg ttgcttagca agtttttact cataactcca agttttgtga 7080
cttatagtga gaaatgggta taattctttg tgatttgtta aaatgcagta tttcgaggag 7140
ggataacata taaaatttaa cagtttccta tcattatttt tatccttttg aaccactgta 7200
tactacagat agacaaaggc agctttggcc acttactcct cagtgtgact aagttgacca 7260
gatgttttcc agaccacagg ttctgtcaag agacagtgtg gagagaaatg ggtaggcagg 7320
tatggaaaga agagtcagaa acagatagtg agctaatctc tacaaatggc tcgttatggc 7380
ctgagctgtg ttctcctccc cgcaaaaaga tatgttggag tcgtaatccc cagtacctca 7440
gaatgtggcc ttatttggag attcgatatt tacagaggta agcaagttaa aatgagatca 7500
ctatagtggg tcctaattca atacgactgg tatccttaca aaaaagggga aatttaacca 7560
cagatacaga cacacacata gggagagtgc tagtgaaggt gaagtcagag attggaatga 7620
tgtagcagaa gcctaggaat gccaaagatt gccagcaaac caccagaagc tgggagagcc 7680
acatggaaca gattctcctt cacagtcccc tgaaagaacc aactctcctg acacctcgat 7740
ttcagacttc tagcctccag agctggggtc aatgagcttc tctcattaag ccatcatcca 7800
gtgtatggta ctttgttaca gcagtcttag caaatgcatg gttcctcact ggaaccatga 7860
atttccatgc atttatttca tttaaataat aaaacggatc ccctttgtgg cttgtagttc 7920
tgttccattt caaaaatcca gaaaaaagat ttgttcagaa gctagaaatg atgaactgga 7980
tcttgcccaa ggtcataaaa ccacaaaacc ctttacagag cacaaaagtc tgatttttca 8040
aagcttctct caaaagatgg gtcactcctt agtcatttag gccactgaca actgccctgg 8100
actctttatt tatttattta tttatttatt tatttaattt ttttgagaca gagtcttgct 8160
ttgtcgccca ggctggagtg cagtggcgtg atctgagctc actgcaagcc ctgcctcccg 8220
ggttgacgcc attctcctgc ctcagcctcc caagtagctg ggactacagg cacccgccac 8280
cacgcccggc taattttttg tattttgttt agtagagaca gggtttcacc atgttagcca 8340
ggatggtctc gaggtcctga ccttgtgatc cacccgcctt ggcctcccaa agtgctggga 8400
ttacaggcgt gagccactgc gcctggcctg gactcttaca tataataagc ttgacttatc 8460
gtacaactta taattgatgc tttcacgtca tgggaagatc aaattaatgc agagagcgac 8520
tacgtttctg gtgggagagc agcaggggtc ttgagaggaa cagcagtgtt gactgtttcg 8580
ctacctactc tgggtgctga ttacatcttt ctttcgtgca ggcaaatcca cttggagcta 8640
ttttgggctc actgtgggca tcattctttg tatactctgc tcctttcccc catagtttct 8700
ctattgatgg gggattacta aagaatgaag aaaagaacaa aatgaaagtg gcttttgaaa 8760
aattttaaat gactgtcact atgtaacata ttcattattt catgtctcca ctggacattt 8820
tggagatgtg agccttgcaa tacctacatg aatgcttcta tgattatgat tattgttttg 8880
ctcttcgcct aacaacttgc caagtattgt caacctcagt gtgtgagatg gggtccactc 8940
aaacatcagg gccgaagtca ggtagttcag ttaagtgaat ttgataccag gaactagtta 9000
caaaggagtt ggaagggctg gaaagccaaa ctggagaaga aggggaaccc cagagtaaca 9060
atagcaggaa gcctctaccg tctacctcta gaactgggga ggagctgagt taacagagtc 9120
ctggagccat tgctggggaa gaagaacccc aactgcagag gaaaagtggc cattgtgaag 9180
aaggtgatgg tggagaagtc gtctgaatca aaggggagag gatatgttgg ctcctttatc 9240
tttttatctt tcattgtcct aattccttct gggcatggtt aaatgagccc aggaaatgca 9300
gttggcagga atcagcttcc tgtgacatag acagagtaag agaaggacaa aaataatgaa 9360
tctgagagca agcaggcaaa tgaccagcac attaagccta gcacacagta tgttatacgg 9420
gatttggggt agcaaaagat gaaggcaggt cagagaagcc acaactaggg gatgggcaag 9480
gtcaatgagg tatgacgggt attgtataca ggaagagggt cataaacagg agtggagtca 9540
atacaggaga ctaacatata cacatcataa atattagtag agaagcataa aatagtctcc 9600
tggagatcag ggaaacagga aattataggt tttcaggata attcagccat atccaggaaa 9660
cacacacagt gaagtaacaa agagtcaata ggcttagagt ggtacaattc attatgcaca 9720
tgtaggaatt cattccaaat aataggctga aatgtagaca tgagaatcca aaaaagatgt 9780
tttcttagtt tttgccaata tcttagctac gttttttttg gttcaacaaa gtaagttaac 9840
agtcatatct gcttggaaat tgtatttagg ccaggtgcag tgtctcacgc ctgtaatccc 9900
agcacttggg gtggctgagg caggaggatc ccttgagccc aggagttaga ggctgcagtg 9960
agcgacaact acaccactgc attccagtct gggtgacaga acaaaaactt attaaaaaaa 10020
gaaaaaaaaa aggtcgggcg cggtggctca cgcctgtaat cccagcactt tgggaggtcg 10080
aggtgggcgg atcacgagtc aagagatgga gaccatcttg gtcaacatgg tgaaacccca 10140
tctctactaa aaatacaaaa attagctggg catggtggca tgcacctgta gtcccaccta 10200
ttcaggaggc tgaggcagga gaatcgcttg aacccgggaa gcggaggttg cagtgagctg 10260
agattgcacc actgcactcc atcctgtcga gactctgtct caaaaagaaa aaaaaggaaa 10320
agaaactatt gaaatagctg atattagttt gcttacttgt cgttactctt tttcatgatg 10380
gattataaag aaaagttata actatttgaa ttttctgctg atttgaagtc tctataaaca 10440
gtacattcct ttttggtaca cagagggcac ttatctgcaa gaaaggcaaa gaaaatggaa 10500
aagttaatga aagaggaatc atccaatcca cgaacagaat gaaaccacat acacagtgaa 10560
gaaacttgtc ttacattttc ttccttatat tacttatcat tcatggtagt gactactttg 10620
gggcttgagt aaagcttctc taatttattc catgtagcat catatgtgaa aaagacaaat 10680
agatacttta gacatgataa taacacttta ttttttattt atttgtttat tttgagacag 10740
agttttgctc ttgttgccca ggctggagtg caatggtgca atctcagctc actgcaacct 10800
ctgcctcctg ggttcaagcg actctcctga ctcagcctcc caagtagctg ggattacagg 10860
cacgcatcac cacgcccagc taattttttg taattttagt agagacaggg tttctccgtg 10920
ttggccgggc tggcctcaaa ctcctgacct caggtgatcc acccaccttg gtctcccgaa 10980
gtgctgggat tataggtgtg agccaccatg cctggcccat aacaccttat ttaaaaataa 11040
tctgtctgga tccatacaac ttgtctggat aactaaattg gaaattattc cttgttttaa 11100
agtaattcaa ttgaaaattt ttaaattttt ttgttaatca agcacttttt ggtggaatct 11160
aaattaacac atgtaggaga tgcctgtttc actaattaca caggcatctt gcagtaatta 11220
atgtctggga ggaaggaatg tcttttgctt actctcttct tcttcacaaa aatgtgaatt 11280
ttggaaagca ataatggaag catgtagaat tatagaaata caaatgtata taactatcac 11340
aaaaaaatga ggccaaagga ctattcagat ataattaggc tatggtagct gtaattatct 11400
aggaaattaa taaaattcat tcacctagaa attattagtg agcatcaaat atgtgtcagc 11460
actaggctag ggtctcagaa cgtacagata aatcatagtt ctggcttcag ggagttatat 11520
agattagaga taaaacctaa ctacaggggc tgggcacggt agctcatacc tgtaatccca 11580
ccactttggg aggccgaggc gggtggatcg cctgaggtca aggagtttga gaccagcctg 11640
gccaacatga taaaatcctg tcactactaa aaatacaaaa gttagccggg aggtagtatg 11700
tgcacctgta atcccagcta ctcgggaggc tgaggcagga gaatctcttg agcctgcggt 11760
ggaggttgtg gtgagctgag atcacgacac tgcactccaa tctgggcgag agagtgagac 11820
cctatctcaa aaccccaaac aaaacaaaca aaaaaaccaa acctaactac agggctatga 11880
gagatgacta ctggcaagga gccacaggta gaacaaaggg gatgtgtccc caggcaaagg 11940
gtagtcagca agcctgcaac ctcagggttc ccggatctga gcctctggct cttggctagg 12000
caggccccaa gcgttggcct cctgccatgg caagctccag cctggtctcc caccttgagc 12060
taacattcat atgttgtaga cacagccacg cttcctgctt acctgtcact tccagttctc 12120
gaaggcaccc ttttcaaatg aaaatccgcc ccttttcaca tcaaacagct catctggtcc 12180
tgtggattac atttctcaga aatgcctctg aacattcgcc tcctctccac ccccactgcc 12240
tctgctacag tgcaggtgct cgccatttct gatttgttct gtcacacact cgtttatcag 12300
gtctctccat ctcctgtttt gacatgctgt aaagcaattt gccactggaa aaaagggctc 12360
tctttttttt tttgagacgg agtctcgctc tgctgcccag gctggagtgc aatggcatga 12420
tctcggctca ctgcaacctc tgcctcccaa gttcaagtga ttctcctgcc tcagcctccc 12480
taaagtctgg gattacaggt gcacgccacc aagcccggct aatttttgta tttttagtag 12540
acgtggggtt tcaccatatt ggccaagctg gtctcgaact cctgacctca ggtgatccac 12600
ccgcctcggc ctcccaaagt gctgggattt caggtgtgag gcatcgtgcc cggcctctct 12660
ttccaaagta tgttattatt tttcccaaga ccctttgacg gatcctcatt tcctacacac 12720
agtggttctc aaacttggat gtactccaga attacatagg aaattcttca tttttttaga 12780
cgaggtcttg ctatgtttct caggttggtc tcaaactctt gacctcgtgt gatcctccgg 12840
ccgcagtctc ccaagtagct gagattatag gcgtgccact gagcccagct ggaaattttt 12900
ttaaaacaca gattcctctg aatcaaaatt tctagaagtg aggtctgagc aatctgtatt 12960
tttaacaagt tatccagatg gctctttact gtcagtctgg ttccagctga gggagtttta 13020
gaattacaag gcaaagtcca aacttagcac acaaagtcat tcacatcaac tcttcccctg 13080
tacacaccct atgtcatagc cagagttgta acccatttct taaacaccca gggttctttt 13140
aagtctctgt gtcattgtat gttattttct ctagaattgc ctttaacctc ctttccaccc 13200
tggaaagcat tccctaagcc atctttgaag ctttctttga tctctaactt agagtcctcc 13260
tctccttcta aagccctgtg ttaatcactt gtcatggtgt actttaattt ttacttgcct 13320
gtttctcttt acggtacttt gacttcaaag ggtggggctg caaattagtc atctccttag 13380
agtccagcct tgttcctagt tcctaactgg cacttaatat aaacgaatga atgaatggac 13440
aaatgaagag aatgctagtt atgataaaga attggccgtg tatgagacta cttctcttta 13500
tgaactaaat aattatatgc ctttcaataa aatactagta cacgtagcta gcacaagctc 13560
atcagcattt gagatgatat ggaaaccaaa ataaacaaat gctaccacaa aaacataatg 13620
actgctttcc ccagtgcagg actgatggaa tcatcaaaca ttgagattaa tgtaatgttt 13680
ggcagatgtc cagtgttttt atttttcatt tgcctttgtg ttcatttatg gactaacaat 13740
ataataaaca cacacatact cacagtacat cttttttttt tttttttttg caaagcccag 13800
ttttcttcat cgcatatctt tgttttcttc aagtatcccc ttacttaatg ttggtagtat 13860
tttttttaat gaaatataaa tccctaacca ccaagcaaga aatgagactc ttaattgcat 13920
cagtttacag tgcaatgtga gtgtaaatag tgtaaattga atttttaatg gacttttttt 13980
tttcccgttt ttgcttgcct tacattcatt atccctccct gggtaataaa catttatttt 14040
tccctttgta accactcctc cccttctgtc catgtggttc tttttagtgg agctggtggt 14100
agggatgtgg catgagattc aagcttggcc acctggagtc actgtgttgt gctccagaag 14160
gacacctgat ccaagtcagc cagtaagagt gagccccagg atttttgctt ggaccactga 14220
ggaaaagtgt actctgccct gtggctgatc aaccattagg atataagcca ttgttgtagc 14280
tgtgtgagtc aagctacctg agaatgagga caacacagtg gcaagcaaca ccaagagaga 14340
aaaagaggca gattctgatg acatttttga gcctttgcat ccagctatgc ctgaatccaa 14400
tttatcccct ggaatttaca cttacttgag ctccacccac ttgaaagaaa acatttcttt 14460
ttattcttag cctgatttga atttggcctc tctcatttac tacccaaagt gtcttgacca 14520
ctagaatatt atgccagact ttacagcatc attgaatttg ccactttcca gaagagttgt 14580
gtgaattttc aatgtagctt ttaccttcta tgagtatcta gagatatatt taagtagaag 14640
tactccacta gtttgttgtg agatcttaag tcacttaatt tctctgtacc ctagttccct 14700
catttgctag acctagggag ctataatgtt ccttctgcaa aattcttatt ttgtgaaata 14760
ttctagaatg tctaactgat acactgctag aacaactgac tgctatttaa gaagagttga 14820
ctgctattta aggatcataa ttctctaggc ataagtgctg tgacggcaca gcgtgtgcat 14880
cggggctgag gggtggggtg gagcagaaag taggaggaga aagtttgata aacttccttt 14940
tggataaatt gaaaacagtc aaataattta taatttctta tattatcatt attagcttct 15000
tctataatta ggagacttgt tccaaaatgt gagaattgtc acaagttgtc aaattcatca 15060
aaggaagaaa atggatgtct cacaaaaaag tatgctcagt ccaatttctt ctcgtcacac 15120
tggaacaaac tgaacagttt tacacagaga tgagaagcct ggacattttt caaatatgtt 15180
ttgaagagaa tggcaatgcc tgagacagaa gtaggaaaaa gcaatgaata tttaaaaatc 15240
tgagctggtg taaaactaga aatagtttta gtaagaacaa tgtgatgtgc tacactaagt 15300
gaaatgtata cattgggcca cattataatc aaaaataaga atgtactttt attcatcttt 15360
tatttaaaca ataatcaagg tggcgggcgc ggtggctcat gcctgtaatc ccagcacttt 15420
gggaggccaa ggtgggtgga tcatgaggtc aggagttcaa gaccagcctg gacaacgtgg 15480
tgaaaccccg tctctactaa aaatacaaaa attagctggg tggggtggca tatgcctgta 15540
ataccagctg ctcggggggc tgaggcagga gaatcgcttg aacttgggaa gtggaggttg 15600
cagtcagcca agattgtgcc attgcgctcc agcctgggtg acagagcaag agactctgtc 15660
tcaaaaggaa aaaagaaaaa aaaaaaatca aggtaccatt tgtaccattt cctggaattt 15720
ctccaaagtg gcaaggtcac atgtttatac attagactcc cagtttaaca cacagcagac 15780
aataactttt tttttttttt tttgagatgg agtctcgctc tgtcacccag gctggagtgc 15840
agtggcacaa tcttggctga ctgcaacctc cgcttcccga gttcaagcga ttctcctgcc 15900
tcagcctccc gagtagctgg gattacaggc atatgccacc atgcccagct aatttttgta 15960
tttttagtgg agatggggtt tcaccacatt gtccaggctg gtctcaaact cctgacctca 16020
taatccagcc acctcagcct cccgaagtgc tggtccaccc ttccttcttt tctcccttcc 16080
atccttctcc cttttattcc atttttctaa atattagacc atagtacaaa tcaaaagtca 16140
caaactgata ggctgcaatg tagatacagc tgggaaaatg ttttgtttgc agaacactgg 16200
ggaaatttaa catgaaaaac tggaagatct caatccacat ggccacatgg taatattatt 16260
aatgttgcag gggctttcca attcaacatg tcctctgcat ccctactatt tatactgcca 16320
ctcatccacc tttctgtatt actggcctat cacctataca cgtttgagtt tatattcttc 16380
tggctttact tagcaactta ccttttatat ttaacattac aacatggtat tatcaattag 16440
tattcgattc agttgcatat aaccaaaaat gccaaattat actggcttaa acactaagga 16500
tttatttttc tgtcatataa aacaagtctg gaggtgggta gtccagggct aatatgacac 16560
tccagtgtca caaggtacct gggctccttc tatttatctt tttttttttt ttttttgaga 16620
cggagtcttg ctctgtagcc caggctggag tgcagtggtg cgatctgggc tcactgcaag 16680
cttcgcctcc cgggttcaca ccattctcct gcctcagcct ccggagtagc tgggactaca 16740
ggcgcccgcc accacgcccg gctaattttt ttgttatttt taatagagac gaggtttaca 16800
ccgtgttagc caggatggtc tcgatctcct gacctcgtga tccactcatc tcggcctccc 16860
aaagtgctgg gattagaggc gtgagccacc gcgcctggcc ctatttatct tttataggac 16920
atggcttcca gtctcaagtt cagtttgtca cccataatgg ccaaagagca gtagtcacct 16980
tacctgtttc aggcaggaag ggcagagggc aagaaacaaa atggtgctcc tcctgattga 17040
gtcagcgttc tttaaagatg ttttccagat gttccaccca gccgcgtttt tttttttttg 17100
agagacaggg tcttgctctt gtcacccagg ctggagtaca gtggcatgat catggctcac 17160
tgaggcctca atctcccagg ctcaagcgat ccttccattt tagcctccca agtagctgga 17220
agtagctggg accacaggca catgccacca taccctgcta actttttcat tttgtgtaga 17280
gacaaggtgt cactgtgttc tccaggctgg tctgcagttt ccaactcctg agtgcaagtg 17340
atcctcctgc tttggcctcc caaagggctg ggattacagg tgtgagccac tgtgtctggc 17400
caagctactt ctacttatat ctcattggtc ataacttgat cacacagcca catccagcta 17460
caatggagat tgagaaatgt agtcttttgg ctgggtacac agcatctgaa taaaatccag 17520
gcattgttac taaggaagaa ggagtaagtg tcaatctctg ctccataact ctctaggtta 17580
atacacacag atggaggaga ttgctagttg cccctcaaga tccagccttg ccttctgggc 17640
taagaaagcc cctgagattt acctggccat agtggcactg ggaacaaaca atgtatttct 17700
aaatcttctg attttaaaat ctttcagaat cacgatttct ccgacatcag tatttttatg 17760
tctttagaat tcaacaaaat gaaattccta agtctaatat atgtgaatat taagttttag 17820
cagatactgc tacataactt tccagaaggg tgtggcaatt cacatctcca ccagtgatca 17880
gcatgttcat tttccataca gctctggata ttttgtctat tttaaaatat cttcttctaa 17940
tctataattt aaaaatgtaa cttagtagga atttaattgt tcatgtaacc aaatcttccc 18000
attaatggct atgggtttct tcttttactt cagaaagtcc tccccacctt cagagtatat 18060
aaacattttt ttctaaattc ccttctaatt tcttataatt tatagtttta tttttgttat 18120
ttgattcatt tattctctca acagatattt attgagcact tattatacgt caggctctct 18180
tcaaactctg gtgagagtat tttctaactg ggagagacaa ccctagttga taagaaacaa 18240
acaaaccaat aagtaaataa gacatttccc ccagataatt aatacttggt gacgggggaa 18300
gacagtgaga caggctactt tacactggca ggtcaggaaa ggcttctctg aggaggcaag 18360
tgattcagga ttaattgatg actggtggag aagtccgggg agtggacaca ggtgggaaca 18420
gcctggtggg tgtgaacagc aacaagaagg ttaatgtggc ttcatggaac agggtgaaga 18480
tgagacaagc tgaacactga ggtgggcacc agaacatgga gggctttgta ggtcccaata 18540
aggagtatgg attttattgt atactggaga tttgtcatcc atctagaatc tgtttttata 18600
tataagaaaa ggtgtatatg tttgcgcaag tgtgtgtgtg tgtgtttggg ggggcggggt 18660
gggggcagac agggcgtaac ttttctttaa attagagtca aaatttaatt aaactattca 18720
ttctttacag gcagtgaggg gattaggatc ttatcccaca gaatctcacc tcatttcaaa 18780
tgttgtacag atattatctg agatatattt tcaggccggg tgcggtggct cacgcctgta 18840
atctcagcac tttgggaagc cgaggcgggt ggatcatttg aggtcaggag ttcaagacca 18900
tcctggccaa catggtgaaa ccccatctct actaaaaata caaaaattag ccgggtgtgg 18960
tggtacacgt ctataatccc agctacttga gaggctgagg caggagaatc gcttgaatcc 19020
aggaggtgga gcttgcagtg agccgagaaa acgccactgc actccagcct gggcgacagg 19080
gcaagactct gtctcaaaaa aaaaaacgaa aaaagaaata tattttcaat gaaatcaagc 19140
atataatgac tatttttatc ccagcatctt ggcttcttca ggctgctata acaaagcatc 19200
cctagtggag catccctaat ctgaaaatcc aaaatccaaa acgttccaaa atctgaaacc 19260
ttctgaacac tgacatgaca ccacaaatgg aaaattctac atgtaaatac acgtaaatac 19320
aaactttgtt tcatgcacaa attaaaaata ttgtataaaa ttaccttcag gctatgcata 19380
taaggcatat atgaaatata aatgaatttt gtgtttaccc atgggtctca tccccaagat 19440
gtttcatttt gtatatgtgc atactcccaa atctgaaaaa atccaaaatc ttaaatatct 19500
gtagtcctaa gcattttaga taagggatat tcaatccctt atccataaac aatttattta 19560
taaacaacac aaatttaggc tttgtaaaaa atagaaattt ggccgggtgt ggtggctcac 19620
gcttgtaatc ccagcacttt gggaggccga ggcaggcaca tcacttgagc tcaggagttt 19680
gagaccagcc tggtcaacat ggtgaaacct tgtctctacc aaaaatacaa aaattagcag 19740
ggcttcgtgg catgcgcctg ttgtcccagc tactcgggag gctgaggcag gagaattgct 19800
tgaacccgga aggcagaggt tgcagtgagc caagattgag ccactgcact ccagcctggg 19860
ggacaagtga gactccacct caaaaaacaa acaatcaaac acaatataaa tttatttctc 19920
atagttctgc aagctgagaa gtccaagatc aagatgtgag cagatccacc tttggtgaag 19980
ggctgctttc catttgatag atggctgtct tcttgtgtcc tcacatggtg gaaagggtga 20040
ggcagttttt tgaggtctct tttaaaagag aactaatccc attcatgtgg gggtctgccc 20100
tcatgaccta atcactttcc aaaggcccca cttcctaata ccatcaccct gggggttaaa 20160
ttttcaacac acaaattggt tggagagtga gcgaacatag acattcagtc tatagccccc 20220
agggatgtag ccactgaata aaataatcta gaacttcatc tagaggcagt ttataagtca 20280
cataagaaaa ccagttttac ttatagtcac ataaaacttt tgataaaaaa caaccctagt 20340
tgataagcca tttgtttact ggacttggct ctgaagaaat gacttttggc taatcctcaa 20400
aatgaaatct actcacagag ggcaagatct gccaccagtg aagatgttta aaacaatgtg 20460
ctgtaggctt tgaaggcaag tctaaagaag agcttctaaa aatattttgc acaatggtag 20520
cattaaaaga gtaggcacat gggccccaag ataacatctg tgaagaggag gaccagtgtg 20580
tgtttccttt tgtatgttta agtactaagt cagtgattac tttataatta aaccatattc 20640
ctcttgatct gtccataata aggtcactct gtaatttata ataggcttct taccaaattc 20700
aagctttaca acaaccctat aggttaaata ttcctgtgaa ttttatagat gaagaaacag 20760
agtaagtgaa agagttcata ctcaattcaa gtctcttgag tctaaaatca gtgctctctc 20820
tacataagaa gttagttgct aagatcacac aacttgagaa gagagaagac agttttcaca 20880
gtccctcccc tctgaccact aggctgcttg gggtactttt acaacttacc tgctctgaat 20940
taaggctctc tgtgaatctg attcatctgc caatttgcaa acagtcagcc ctgtagtacg 21000
tacctaagtt gatttttaaa atactatttc tcatcttaaa ataatggaaa ttgagtgaca 21060
taaatttcct ggacactttt taagatccaa agacaatttg ctaatttgtc gttacaatta 21120
aagagccccc caaaaggcgt aagaactgga ttttgacttg gacttctgtt ccttagtttc 21180
tttaacatat tgggggccgg gagtggtggc tcacacctgt aatcccagca ctttgggagg 21240
ccgaggcggg cagatcacga ggtcaggaga ttgagaccat cctggctaac acagtgaaac 21300
cccgtctcta caaaaaatac aaaaaaaaaa aaaaaaaatt agccgggtat ggtggcgggc 21360
gcctgtagtc ccagctactc aggaggctga ggtaggagaa tggcatgaac ccaggaggca 21420
gagcttgcag tgagccgaga tcgcactaca ctgcactcca gcctgggcga cagagcaaga 21480
ttccatctca aaaaaaaaaa gaaaaagata ttgggaaaac tcacttaaac tctatgggct 21540
tctgtaccac taaggcttct caaatttgat gcaagtaatg ggaacttgct gtggctaact 21600
tgagccacag gagaaggata ctgcaagact cacaaatgct tgaaagtttc ggaacaggtt 21660
tggaaatggg taagaaccaa ggaccgggtt gacctgaact gaaaggaaat tgaggtgcta 21720
ttagggacaa atatgaaatg aaggatgaac tgtctctgac atcctttgct tctgtgtttg 21780
gagatgagta agcagggagt gggtgcatat gcaaacaggg tggtcagaaa tgctttggcc 21840
ggatgtgtga ctgaaggcta tgtgcagctg aggaaggctg ggccaccaca gtagcagaga 21900
ggctgaccaa tttggcatgc agcaagcagg aagtagaagg gatcttaagc aagcaaagtg 21960
tttgggtaga caagattatg aggaatgagc accaatgaca tttccaacaa gcactgaaag 22020
cttccaaatg cttctaacag ttactgcctg tacaaacaca ggacagtctt gactatgtga 22080
gactgtgaca gtttcctcat gcctatgaaa tgagagacag tacttagcac tgacatgagc 22140
ttcatatttg aaggcagcaa actactgaac cacatgtagc ttccgaaatc aaaagatgca 22200
aactggcaga ccacaggtca gatatgacat gcagaaaaaa attagttact atcttagcct 22260
agtttgtgct gcttacagaa tatcagagac tgggtacttt atagagaaca caaattattt 22320
ttcatagttt tagaggctgg gaagtccaag atcaaagggc catcatctcg tgaaggcgtc 22380
cctgctgtgt caaaacatgg tggaaggcat cacatgggca aaagagagag agggaagcgg 22440
gggcaaactc ctttttatca ggcacccagt cctgtgattc attcatgaag acagtgtcct 22500
cctggcctaa tcacctctta aagctcccac ctctcagcac tgttgcactg gaaattatat 22560
ttctcacaca tgaactttgg ggaacacatt caaaccatta cagttaccaa agataaaaat 22620
gcagatttca gatttctctt ggggaagaaa aaaggctctg gcaataccag ggctgtattt 22680
ccacatggca acagttgtct gttgccttct gttgggttgt gggctctcac attgccatac 22740
cctcctcatg gcccccttca ctcataacag tacctgccta gacctcgaag cactagtttc 22800
caaccctgct tgagttaact acttccatga caactgtgca aggccagagt gtgggcagct 22860
accttgaggg ccaccggcat gggctgccca agtgaccgag ggcactaacc tctgctctcg 22920
aaacttcttt ggtaggatct cttcactcct gacttactac ttgtaactgc tggaaacaac 22980
catttattct acccctacta aaccacatct gtcttttctg tcagagccaa tcttcaagca 23040
tatgcttcct caaaagactt taattaccag gtagtagaag cggcagcaaa gagtccccat 23100
ctagcatctt ctaaaatgta gtggagaaac caacgacaac gaatctgaga caaccagttg 23160
tttgacttct ctgaaaacaa cctctaaaaa aagccttctc tcgtgaatag ctgcagacat 23220
gctgggcact aattgatgat gatgcccttt agtgagtctc ctctgcacat acctgattcc 23280
atttggatgc tggccctcac atacttctat ctgcgaactc cggaacacag aatatcatac 23340
aatgtatgat tcaatctgga ctctgctgga gccattctct acaaagcaga cacaactttt 23400
tcttgcctaa atcctttcaa gttgctttat ttttattttt aaactaatta ccataaatct 23460
aatttatcaa gctgctttta agataaactt taaaatgctt agagcagtct acaaattctt 23520
gcactgtttc actcttcctt atctctcagt cttcatctcc cattgcatct tctctgactc 23580
tttcatatgg atcttctttc aatttgtgga agaagccaag cttgttcctg cctcaggtcc 23640
ttgacacttg ctgttcactc tcctgggaga ctcttccccc catcttagct tttaaatacc 23700
agctggtcag actccttacc tgccttccac cttcctctca ttctcccact cctcgccttc 23760
atttctcaga gtcctccgct ctcttctttc tattgctctt aacacaattt gtctgttttc 23820
cgctaatctg taaactctac aagagcactt aactattttg catcccaccg aatattcgga 23880
gtcagcacag accccgtcat aatagagtag gtaagcaatg cttacctgtc aaatgaatga 23940
atgaaacacc cgattcatcc cccaagccag cttaatacat tagatgatct tatggactga 24000
ataagacatc cctcctttta aagaatattt ttgttttagc ttcacatcag caaaccccga 24060
acggttcatc ctaagataag cctggtagag aggaaagata ctaaccttct agggtcctaa 24120
agtggaacta ggaacttatt agatatgcac attctcaggc attatttccg atagcccagt 24180
cagagaattc tgggggtatg gcccagcaat ctgttttaac aaacctttca ggtaattctg 24240
atacacacta aagtttgaaa ggtttcttta aggctttcca gttttctttt cttttttgga 24300
gggatggggg ggggtctccc catgttgccc aggctggtct cgaactccta gcctcaagct 24360
atcctatcct cccacctcgg ccttccaaag cattgaaatt acaggcatga cccactgcgg 24420
ctcgcctccg tttctaactt aaaaaaattt tctttggagg ggagccttaa tcctttctat 24480
tatcctgcta gtagaatctt actactcctg agggtaatct ttgtcataat ccaaaagcct 24540
tgaataaagg cctatttgta tagttatgca gaatatattc ctgggaggtt tgccttccta 24600
ggagcacagg tcggccctgg gaggtggggg ttggggggca gggctgcatt ctaaagtcct 24660
gtaacaacgc acaagtacaa ctgaatagaa ctcggaacaa aggcatcagg gccacggtgc 24720
aagtctttgt tcatccgttc cccgactgct caccctgtct gataccgctc ttttccaccc 24780
agaaaagcag ccactcaagt tttaagaatg gatgtatgcc gcggacgttt tcgattaggc 24840
cgttttctct caggcactgg aggatattcg tggctgcagg aggcgcttcc accctttcct 24900
caacctagca aagaagtaac tgaaactaac ccaagggtta caaccgaaaa gccccttcca 24960
gcttcagaag cagaactgga agctcggata gacttctccg cctctacact cctggaaaac 25020
ccgcagtgga tttcaccaac ttcaggatcg gagcccaggc aggcgaacgt accttattgc 25080
gcatgctcgc tagcccctcc cgctcggagc ggaaggggga gcgctggggg cctgggcctg 25140
gcctggccgg ggcgtcggca ccggcggcca tcttggcttc ccggggaaag gcggcgtgag 25200
gggaagaagt tgtagggtgg gggcaggagt gaggaggagg gaagagagag ggggaggagg 25260
ccgcggcggg gcagggcggg gactgcctgc ctgcctgggt tgcggaagtg atagccgccg 25320
accgagcctg ctgctttctt gctactgctt cggcttcccg gctacccccc ggacggtgaa 25380
ggcggcccag ctgtggatgg tcagatagcc cttgtctccc gccgccaatc tctggcccct 25440
agcagcacgg agcagacggc ggcagcagca gcagcaggcg aggaggaaga tggcgggacg 25500
gctgccggcc tgtgtggtgg actgtggcac ggggtaaggg ggcttacggg cgggggtggg 25560
gaaactgagg cggaggaagg aagatggcgg gggagggagg aggccgggaa atgaatggtg 25620
cggcgaggtg ccgccgccgg ctgtcagtcc tagacccgcc ggccagcgag gggtggggcc 25680
cgcagccagg gcctcgcggg tcccctcgtt tctccctcct gggactgggg cgggggcgcg 25740
ggcccgagat tcaaccccca accctcccag cggctttctc cgcgcgaccc ctcccggccc 25800
ttcccccact acggtgggca gcgccgccca aagggcgctg gggacggtcg tcttgggggt 25860
ggtccccggg cccgacccat ccggctttcc tttccctccg cgcccgtttt tgccagtcgg 25920
tttggggacc caggggccga gctcggggac tggcctggca ggggagctag aaaagagaag 25980
cgctcctggt aggtttgaca agatcgctgt gacaacattc tgcccagggt gtgggtgggg 26040
aaagggaaga atcggactct gaaaatggga acctacagtg gggctttcat ttgaccaccc 26100
accttctcct ttcgactcct ggtcatttcc atctccctct gcgttttaac cggtgaacca 26160
agtcacattt taattcgagg gagaaagatg tcatgtgtta cttctgtagc ctcaaaaaag 26220
tcccccagtg agcaagcgcg ctgcaacttc cttagttttg tcaaagccgc ttcctgcttt 26280
cagtctttta tacccttata aggtagtttt tagtttccaa cctgagcaca tcttactaga 26340
acttttaagc aggttttaaa gagactgaat taccggtgct tggtgtccat ttatatgact 26400
taaaaaaatg tacttatgtt tcatggagtg ggggaaagga agcaccctgg aaaataaact 26460
aatttaagtt gtattcgttc attctgtgat ggtatcttga aggaagcagg aaggtataga 26520
ggtatatagg agggtgttta agttgcaaat agttactcgt agatatgtag ggtagtttgc 26580
gaaaatgtag agtggcttta aagtgtggtc gttcattttg tattaataga cgttttagag 26640
agttgtacca cactcataac tgcatagaga atagactacc cttattttta tgtagaatca 26700
ctgcttcagg atgacatata ggaaagtggt ttttttttct gtgctgatga catctctcca 26760
attcccagaa cactcccagt tttttttttt tgagggggaa gtc 26803
<210> 19
<211> 26803
<212> DNA
<213> Homo sapiens
<400> 19
gccgcaccta ccgctggcct cagacatcag caccccaaag ggtatgttgg agtcccatgg 60
tggaggtgcc ggccgctcct ccacgcactt gatgtatacc gaggatccca tgcttgacgt 120
cgcagggggc attagccagg agcacgttgc tgggatgcca gtccaggctg aggatggtgg 180
agccaatggg cttcttgatg tacttgcaca cccaccagtt attctcccgc tggaaataac 240
agatggcgat gacacacagc tgccgcccac agcaaacttg tcctgtgggc ccagcacaca 300
cagcaggcag tgggtgcggg cttaccctcc tggcccgtca gcgtccacac agaggccttg 360
tggcctgtgc cataggtcac gatgaggtta ctcttggagg cccagttgat gccttttacc 420
tgcccgttgt gctccttgag ctcgggcacc ttggcccact tggccccact cttacagctt 480
cgtggttgtt gaggcagatg gtggccatct gggtgcggcc cttgctccag gtttggcagc 540
aatgggttcc tgcagtcagc tgtgggaggc tgtagttgct ctccttttca gagtacccca 600
gctgcagact ctgggcagga cgcacccaac cagtgctctg gttttttgaa gtaaaaaatg 660
cataatgcaa ttttaccatc ttaaccattt tttaattatg aaatccggta gtgttaagta 720
taattgtgtt gttgtgaaac agatctccac aactttttca ttttgcaaaa ctaaataact 780
cctctttccc cctactccca agctccaggc agctaccatt tctgtttcta tgaatttgac 840
tacttatatt acctcatata aaggggttca taagtatttg tcttttcttg cttggttaca 900
tcacttagca tgatttctta agtttcattc atgttgtagc acatgtcaag atttcctttt 960
ttttttttaa aaaaaaggat gaagactatt cgtttgtatg tatatgccac attttgttta 1020
tccatccatt tttcttcaaa tgtttgagtt gtgtgatgtt taattatgtg tcaccttgac 1080
ggggcaaagg gttgcccaga cagctggtaa aacattattt ctgggtatgt tagtgaggct 1140
gtttccagaa gagattagca tttgaattgg caggctgaat aatgaagatc tgccctcatc 1200
aatgtggatg ggccagaaca aaccaaaaag gtggagaaag gaccagttat ctccccacca 1260
cccctgtgcc ctttagctgg gacatccatc ttctgccatc agacatcaga gcttctggtt 1320
ctcaggcctt cagactccag aagttatacc agtggcttcc ctggcttttg gactcagact 1380
ggggtttcac tgtatgtttc cctgattctc aggcctttgg acttgaattg aattacagca 1440
tgaactttcc tagttctcta gcttgcatat ggcatattgt gggacttctt gacctccata 1500
atcatgtgag ctaattccca taataaatct cctcttatgt atctataatt catatagata 1560
tgatatctat atatcaatcc catagataaa tagatgtctc atatatatat caatgtctct 1620
atatgtagat gtctcatata tggtatctat gtatctgtat catctctata tatctaatca 1680
tctaatatag atgtctcatg atatctatat agatatatct atatcataga tgcagagata 1740
tcatatatat gtctgtttct ttggagaatt ctgactgata caggttgctt ccgccttttg 1800
gctatcatga gtaatgccgc tgtaaccatg ggtatgcaaa tacttctttg agaccctgct 1860
ctgagttctt ttgggtaaat acccagaagt ggattgctgg atgatttggt ggttctactt 1920
ttaatttttt gaggaaatgc catactgttt tccataatgg ttgcaccatt ttataatctc 1980
accaagagtg cacagggttt caatttctct acatccatac caacacttat tagtgtgtgt 2040
gtgtgtgtgt gtgtgtgttt aatggctgtc ctaatgggtg tgaggtgaca tttcactgtg 2100
gttctgattt gcacatctct gataattgct ggtgttgagc atccttccat atgcttgttg 2160
gtcatttata tatcatcttt ggaaaaaatg tctattcaag tcttttgtct attttttttc 2220
cttccaactt ttattttagg tctgggggta catgtgcagg tttgttacat ggataaattg 2280
tgtgtcatgg gggtttggtg tacagattat ttggtcgcct aggtaaatga gcatagtacc 2340
tgataggtag ttttttgacc ctcccccttc tcccaccctc caccctgaag taggccccag 2400
tgtttattgt tcccttctta gcatctgtgt gtagtcaatg tttagctccc acttataagt 2460
gagaaaatgt ggtatttggt tttctgttct tacattaact tgcttagaat gatggcctcc 2520
agcagcatcc atgttgctgc aaaggacatg atttcgttct tcttatagct gtggtgtata 2580
tgtaccacat tttctttacc cagtccactg ctgatgggca tctaggttga ttccatgtct 2640
ttgatattgt gaatagtgct atgatgaaca tatgtgtgca tgtgtctttg tggcagaaca 2700
atttacattt ctttgggtat atatccagta atggggttgc taggtggaat gattaattca 2760
cttttaagtt ctttaagaaa tctctaaacc tctttcccca gtggctgaac taacttacat 2820
tcccaccagc agtgtccaag tgtttccttt tctgcacaac cttactaaca tctgttattt 2880
tttgactttt taatgatagc cattctgact catatgagat cgtatctctc tgtggttttg 2940
acttgcattt ctctgatgat tagtgatgta gagcattttt tcaaatgctt gttggccgca 3000
tgtgtcttct tttgagaagt gtctgctcct gtcatttgcc cactttttaa tgggcttgtt 3060
tgttttttgc ttgttaattt aagtttcatg tagattctgg atattagacc tttgtcaaat 3120
gcatagtttg tgaatatttt ctcccattcc ataggttgtt tactctgttg atagtttctt 3180
ttgctatgca gaagctcttt agtttagtta ggtactactt gtcaattttt gtttttgttg 3240
ctttgtccat tttaaaaaat agagtaattt gattattttg ttgttgaatt gtaggaattc 3300
tttatatatt atggatgcta acatcttatc aaatatgatt tgcaaaacat tttccctcat 3360
tctgtaggtt gccttttcac tattgtgtcc ttcaatgaac aaaagttttt atgtttgatg 3420
cagtcccatt tgtccatttt ttttggttgc ctgtgatttt ggtgtcatat ccaagaatgt 3480
ggaatgtgtc atatccacat tcaatgtcct gacgattttt ttctatgttt tcttccagaa 3540
gttttattgc tttgggtctt tggtttaagt ctttagtcca ttttgagtta atttttgtat 3600
gtggtgtaag aaacaggtcc aactgcattg ttttgcatgt aaatatccag ttttcccagc 3660
atcatttgtt gaccagactg tccttttccc atttagtgct gttggagctc ttcttggagg 3720
tcagttggcc atgtgtacac aagtttattt ctgggctctt tattctgttc tattgatttg 3780
tatatctgtc tttattccag taccacactg ttttattctt ctttttcaaa attgtttgac 3840
tcttagggcc ctttgagatt ccatatcaat tttaggatga attcttctat ttctgcaaca 3900
aatgctattg gaattttgat aaggattgaa ttgaatctgt agactgtttt gagtgatatt 3960
aacatcttaa taatattaag tctaatccat gaatgtgaga tgtttctatt tatttatctc 4020
ttctttgatt tctttcagta atgttttata gttttcatta tataagtttt tacttcctta 4080
gtcaatttct aagaattgtt ttctttttga tgccattgca acgggaatca ttttcttaat 4140
tttttcagat gctttcacta tttgtgtata gaaatgtaac tgattttttg tatgtgtgtt 4200
gattttgtat ctggtaactt tactgaattt ttaaatttgt tctaacctgt tttttttttt 4260
tggtggaatc tttaggattt tctgcatata agatcatgcc atctacaaac agaaattttt 4320
acttctttct tcccaatttg gatgcctttt attgcttttt cttgtctaat tgctttggac 4380
tggagttcca atactctgtt gaatagaagt gtccagaaca gacatttttg ccttgttctt 4440
aatcttagag gaaaagcttc cagtttttca ctgagtatgc cgttagctgt ggacttttcc 4500
taaacaacct ttattatgtg caggtaattt ccttctcttt ccacttttga gtgttttttt 4560
tcttttcttt tctttttttg tgtgtgaaag gatattgaat tttgccaaat gcttttcctg 4620
catcagtgga gatggtcatg tgggttttgt cctttattct gtaaatgtgg tgtactacat 4680
tgattttcat atgttgaacc atccttgcat cccagggata aatcccactt gatcatggtg 4740
aatgatcttt tgagtgtgct gttgaattta ttttgctagt atttttttga ctacgttcat 4800
cagacatatt gggtaattat ttattttttc ttgtagtatt tttgcctagc tttgatatca 4860
cactaatgct gcccctcaaa gaatgacctt ggaagtattc ctttctcttc agtttttggg 4920
ggattgatgt aaattctttt tttttttttt ttggagacag ggtctcactg tcacccaggc 4980
tggagtgcag cggcacaatc atagctcact gcagcctcta actcctgggc tcaagcaatt 5040
ctgcttcagc ctcctgagta gctgggacta taagcatgtg ccaccatgca cagctaattt 5100
atatatatat aatataatag ataaatatat atatatatat atatatatat atatattttt 5160
tttttttttt tttttttttt tttttttttt tgcagagaca aggtaggtct tgctatgttg 5220
cccaggctgg tctcagactc ctaggctcaa gtgaacctct cacctcagtc tcccaaagtt 5280
ctgggattac aggcatgagc caccacgtct ggccagtgtt aattcttcaa tatttggtag 5340
aattcttcag tgaagtcttc tagtcctggg cttttctttg ttgggaggtt tttaattacc 5400
aactcaattt ccttactagt tattggtcta tttatatctt ctatttcttc atgattcagt 5460
cttggcaggt ttggtgtttc tagaaattta tccatttctt ataggtcatc cagtttgttg 5520
gtatagttca tagtcttctc ttataatcct ttcttatctg tagctcagta cctcctatct 5580
ggagatgcag aattaggaat tgaaactata ataggccccc cttttataat ggggtgtcct 5640
tatttttacc ttctcacctt gcaataccct tgtcttgcta gtaaagaaag aagggaaatt 5700
tgattcagga ggtaatcata cttttcaatt tgtacaagat ctaatagcca tgaactccta 5760
tgtcattcct caccatccca taattcttag cccagccatc atccttacct caatccctgc 5820
tggtgcagcc tggtttattc tgttaatgtg ctgtactgca ttgaggtaag gacttctgct 5880
cagctttctt cttgatacca ttgtaccttg attcacactt cctcttgtga aataaagttt 5940
agcatgaagc tgctttctta catattttaa gttcggctta aaggtttttc tgtacatcgt 6000
gaactgtaac aagtggaata taaccagacc gtagcttaca cttgtgccat ttaccaagtt 6060
ttggccaatc aaatgtagcc aactgtttga actgtattca aataagggaa atgctcagct 6120
gtaaccaagc caactgtttc tgtacctcac ttctgttttc tgtatgtcac tttccttttt 6180
ctgtccataa atcatcttcc atggcgtagg tgtgctggag tctcagagtc tattctggct 6240
caggaggctg cctgattttg aatcattcat ggctcaatta aacttcttta attttttttt 6300
tttttgagat tgagtttcac ccttgttgcc caagttggag tgcaatggtg cagtctctgc 6360
tcactgcagc ctctgcctcc tgggttcaag cgattctcct gcctcagcct cctgagtagc 6420
tgagattaca ggcacatgcc accatgccca gctaattttt ctatttttag tagagacagg 6480
gtttcgccat gttggccagg ctggtctcga actcctgacc tcagcctccc aaagtgctga 6540
gattacagat gtgagccact gcacctggct tcactccttc aaatttaatt cagctaaagt 6600
ttttattttt ttctctttct tttttttttt tttttttgag acagagtcac tgtcacccag 6660
gctgaagtgc agtggcacaa tctcggctca ctgcaacctc cgcctcccag actcaagcga 6720
tttctggcta atttttgtgt ttttagtaga gatgggtttt caccatattg gccaggctga 6780
tcttgaactc ctgacctcag gtgatctgcc tgccttggcc tcccaaagtg ctaggattac 6840
aggcatgagc cactgtgccc agcctcaaat ttaagaagaa acagttaaca tggacttgca 6900
tacctccagg atattgtgaa tacctctcaa tatttttcta agttctcaaa gtcaaattag 6960
actctatgac aatcactcaa ggttcccttt gtttccaata tgttgatgac ctgctacttt 7020
gcaaccaaag caaacagggt gctcttctgg actcccttac tcaccttaag gcactgactg 7080
actaaggtta taaatcttcc aggtccaaat gccaacaggt acaaaaaatt cttacctact 7140
taggccataa aatattttag ggtactcaaa aactcgtccc aaaatgcctt gaatcaattt 7200
tattcattat ctctcaaaga caaacaatta cgtgaatttt taggagcaac tggatattgc 7260
caatggattc ccaattttgc tgcccatgtc taacctttat atgctgtcct cttagataca 7320
accacagagc actttacctg gtactctgag gcaccagcct ccttggaagc attgtccaca 7380
ccccagccct tcgactaccc aactttgaca atctttttta cctatattac tgtgaaaatg 7440
atgggattgt tgtgggtatc ttaggacaat ctttttgtcc cataacatat ttctcatgtc 7500
aacctagata tgtcccacag tacctttctc atgttaagta gcatcaggca ttcctccatg 7560
cttatatgca atagacttag ctgccatcct aattgacaaa gcaagtattc ttatactgcc 7620
ccaccattca cctctctgtt ctccatgctg ttcctctaca ttggtatccc tttcaaagtg 7680
gatcttgcct caactaaccc ttaactgttt tttttttgtt tttgtttttt gttttttgac 7740
aattctggca ctaccccaac acctgcttgt tccaatcttt caaagcctgg ccaccttaaa 7800
tagttaatct ggttactggt taggttaaca tctacataat gcagaagagt ttttgttcct 7860
accaatgtaa atatacctgg tgaacccatt ctggacaatg gatatataaa agcatatggt 7920
atccataagc aggaagacaa agtcactctg tctcctcctc tggtagatta atttggcact 7980
gggaagcctc catggaagct caaggtcagt gctttaccca agtaagacta ttggaaggaa 8040
atctctctac attgaaaata aatacagtac tggacctttt ctgggtaaca tttcaaaaat 8100
gtattgcaat caaattctgt gatttggctt cacagatggc actaaacaac cctccactgt 8160
cattgttact tggattataa ctaagtattt caatgtgagt gaatgtcaga ttacaggata 8220
atattcctgg tttgtaggaa aaagtagtgg gatatggaat agctcaagtg ttcccctagt 8280
tggcttgacc agcacccatg actatagttg gatggatgga ccttaacttg gtcaataaat 8340
aaaatacatt tctatagtaa ccaaggctaa attaaagggt ttctctgcca ggtgtttcac 8400
aacctgtttt accctcatag cctaacagaa gcacattgga agtgaagatg tgcagatgcc 8460
aacatgattg atgataaggg ttgtgaaaga cacagaattc caacttggtt gttcacaggt 8520
tccaccacta tgaccttgtc tgtaaacaac actggtctct ctctcttttt tttttttttt 8580
agaaggaatc tcactctgtt acccaggctg gagggcagtg gcacaatctc agctcactgc 8640
accctccacc ttccggttca agcgattctc ctgcctcagt ctcccgagta gctgggagta 8700
caggtgccca ccaccatgcc cggcttattt ttgtagtttt agtagagaca ggatttctcc 8760
atgttggcca ggctggtctt cacctcaagt gatccgccag cctcggcctc ccaaagtgct 8820
gggattacaa gcgtgagcca ctgtgcctgg cctggtctca tttttatgta gcaataagat 8880
atataaaagg ttcccaccta agtggtcaga gaaatgtgaa gttggatatc tggtgccttc 8940
ccttaccaga tatcccactt tgaatgctag ccacattaca actggggttc ttttatatat 9000
aaattaatgc catgtagatg cacctgatga gacatcatag acaacgcact tatgtattgc 9060
aaccctaagt tctcagtaat gagaatgctt ttccaaagcc tggcaatgta cgatgtagaa 9120
agaacaatcc taagcattcc aaagtaatga acaagcatct ggtgccacta tacagactgg 9180
gaagcctgcc atttacaagt tagcagtgta gcctctgttg cactccaaaa ttgtgtccta 9240
gatatgctga ctgcccaaca gagaggagct tgtacaatca ctggcaacaa tgctacttct 9300
atataaatta gaaaggaaaa attgtgtcta atctatatca tttaaaaaga aaggttggcc 9360
gggcacggtg gctcacgcct gtaatcacag cacttcggga ggccgaagcg ggcggatcat 9420
gaggtcaggg gatcgagacc atcctagcta acatggtgaa accctgtctc tactaaaaat 9480
acaaaaaatt agccaggcat ggtggcgggc acctgtagtc ccagctactt gggaggctga 9540
ggcaggagaa tggcgtgaac atgggaggcg gagcttgcag tgagcagaga tcacaccact 9600
gcaccccagc ctgggcgaca gaacgagact ccgtctcaaa aaaaaaaaaa aaaaaaaaaa 9660
aaaaaaaaaa gaaaggtcaa cattctacgt taggtaaata aggcatagtc caattaactg 9720
aactgaactg acctattccc acgactggga gactggttca acagaatatg gaccagtgtg 9780
ttcagatttt tctctgttgt aatctatgtt cttctttcat tttgcagatc tcttacctcc 9840
cagcacctaa tatgagtctt ttctacatag gaaataatcc aaatatttgt gaaacttcat 9900
gtgaagtttc aaatggggga agtgaaggaa cgaacaaccc acctcctaac cccaatttat 9960
acccacctgt gttagttcat ttgtattgct atgaagagat acctggaggc tgggaaattt 10020
ataaagaaaa gaggcttaat tggctcacag ttctgcaggg tgtataggca tggcatcagt 10080
atgtgctcag ctcctggtga gggcttcagg gagcttacaa tcatagctga aggtgaaggg 10140
ggagcatgca tcttacatgg tgatagaggg agcaagagag agggtcgggg ggtgccaggc 10200
tctttaaaca accagctctc atgtgaaata ccagagccag aactcactca tcaccatggg 10260
gatggcacta agccattcat gagggatctg cctcatcacc caaacacttc ccagtaggcc 10320
ccacctccaa cttggggatt gtatttcaac atgacatttt gaggggacag atatccaata 10380
ttcaaaccat atcaccacca gaaaggcata aagattactt taaactgaaa atattggaac 10440
aaacaatcgc tgcggaaagc agccttatct gaccgaaagc agacctgtcc aagagttctg 10500
ctatcatcaa ctccttctga gggactttcc agacagcgag gtgacagatg tttacaaacg 10560
tgacgttaaa aaataaagtt gtgtaaatga gtcttatcag aaccttttat actttctcac 10620
tgaagcctca gttggtatat aaacccttac cactggctgt ttgggaagtg actccttaca 10680
gagtgctccc tcaggcatgg agaataaact tttctcctgt taatgtattt tcaactaatt 10740
cgcaggccct agaccactca gatctaagtt gacagatgaa atgttttcct cccaacatga 10800
ccaagcttat agtgctttta tggtatatct gtagagattg cggtcaggca taacattgtt 10860
tgttagggtt gcagtacctt tcaggaaatg ctcaaagttt aattgattta ggggactttc 10920
actggatgag atgcctctct gagtcatcat aggttgcaaa gtattagttt tttgccactc 10980
actgtgtgtt gtgatatgac atactcataa tggggtagga caagatgtca gttttgcttc 11040
caaactttcc ttacttgttt cctttttggt tcaccactaa tgttgaagaa atgcaagatg 11100
gaaaatctca atagttgaca ctgattgatt gattgagatg gagtctcact ctgtcacctg 11160
ggctggagtg cagtggcgtg atatcggctc actgcaacct ccacctccca ggttcaagca 11220
gttctcctgc ctcagcctcc cgaacagctg ggattacagg tgcccgccac tactctcagc 11280
taattttttg tatttttagt agagatgggg tttcaccatg ttggccagac tagtctcgaa 11340
ttcctaacct tgtgattcgc ccgcctcggc ctcccaaggt gctgggatta caggcatgag 11400
tagttgacat ttattaagtg tttatatgtg ataagcactg ttagattatc ccatgtaata 11460
catcacttga ttgatttaag agattgtcat ttgaaagatt aaagaatatt tcccagaaag 11520
gatgcttggt caagcctcat catcctggga tggatgggac cttggttttg ggcaagttta 11580
ttacaattta agactattct aggtcatgga agtgtgtgtt aggacatata agtagttttc 11640
ttgtccagaa tactttgtaa taatataccc taagtttgat cagctttttg gcattcactt 11700
aattaaaatg ttcacaagta ggtacatttt tagaattgtt aagtaatata ctatttgtag 11760
tagcaaaaca ctacaaacaa atacctaata taataagagg gagtggttga ataaactagg 11820
gcatgtcaac taaaaaacat atttgaaatg cagggaaaat gcttactcta ggcaatagtt 11880
acaacttttg tccattatag gtacataaag gaaaagagta cacaaagaat tctattaatg 11940
atgaaattag gaacaatgtt cccatcccca atttataccc aaaagaaaat atgttctggc 12000
tgggcgcggt ggctcatgcc tgtaatccta gcagtttggg aggctgagac aggtggatcg 12060
cctgagctca ggagttcaaa accagtctgg gcaacatggc aaaaccccgt ctccaccaaa 12120
aatacaaaaa attagccagg cgtgatggca catgcctgta ggtccagcta cttgggaggc 12180
cgaggtggga ggatcgcttg agctcgggat gtggaggctg cagtgagcaa agctcacacc 12240
actgcactct agcatgggtg acagagtgag accccgtctc aaataaataa ataaataaat 12300
gaaaacttat attctataac atgtattctt atgtctttgg ctaaaacaaa agagaagaaa 12360
aataagtaga gagctaagtg aagtgaagtg tcttgaaaca gataagccat aaagtgagat 12420
atatgagtaa atgagtagtt gctggcctag gaattgtttc tgatgttttc acctattgaa 12480
agaatttttt tctttttgta aatctttttt ttggtttgtt aaactgaaaa ataaaagcaa 12540
tgaagagata atattgctgt agtaatgtcc cttagtgcat ccttggagtt agttgtcttg 12600
aatttggcct gctctgattg tcatttgctg tttgctgcat tgaaaaaatt tcagatttac 12660
aaaaatatgt gtgccagggt ggaggaaatt tttaacttga tgaattctca cctttaggac 12720
actgtctgct cttatatggc acttagcaag atgctgcaac aattgtccct gctcatactg 12780
ttggaggaag agggtatttg cagggagatt gctggagtca agatgatcca aatttaaatt 12840
gcagtggtga gcagtggcca ctagagggct gtgaagtact taattctaaa atttccacat 12900
tttaatctgg agcagaggaa actctggaag gaccactggg actaagggag ctcaatccca 12960
aacatcttta tctgagccct aaatgtcttc tacgttattc tttgacaata ctttgttaca 13020
tctcagaatt tgtagttttg aggtggaggt ttaagttaga aatagtttaa tatattctat 13080
tccagaatat aattcgcttt tttccaaatg aaactggcac taaaagcttt ttacttgaaa 13140
gcaaatacac ttcatatgct ttatgtattt ttgtccctat atcattgaaa tagtagattc 13200
aaggctgttt gtaaaaataa gtgactatac tgcatatagg aatagcccca ccttaaagac 13260
tttgatttta aggacattaa gtgggagggt cttttttgcc tttttttttt tgactatcaa 13320
aatactttca catttcttca attcaaagaa caactgttct aaatttagaa gacagacaag 13380
tgaaagagaa tatgctgcca tttccgatgg ttctttttta ctcttgatcc atcactctcc 13440
tgtctttggc tgactctttc tccaaaaagt accccttatt tttcctatgc ctttaacttc 13500
tgttttactt actctattgt agaactttga gaagttgtac accacattca ctgctacctc 13560
acattaactc tgcttactca ttgcagtctg ccttctgctc ctcaatgatt gctcctgtga 13620
aggtcatctt tggtttagca cttgtttgaa atctaatagc ctaaatgcac atcgattact 13680
gtttagaatt caaacgacat ttatttattt actttgagat ggagtttcat tcttgtcgcc 13740
cagctggagt acaatggcat gatcttggct cactgcaacc tccacctccc aggttcaagc 13800
gattctcctg cctcagcctc ctgagtagct gggattacag gcaccctcaa ccacgcctgg 13860
ctaagttttg tatttttttt agtagagatg gggtttcgcc atcttggcca ggctggtctc 13920
gaactcctga cctcaggtga tctgcctgcc tcagccccac aaagtgctgg gattacaagc 13980
gtgagccacc acgcccggcc caaatgacat ttatttttaa aagcaactaa accaggaaag 14040
accttcactc attcaatcaa aaaacatgca ttgaacacgt attcttggct agatccggtg 14100
ctgcaaagat gaaagacgcc actcctagcc tcaaggagct tgcagtttag tggtggcact 14160
ggatgtaaaa taatcataag atgtgaggtg aagactgatg aggtctattt tgttgacaca 14220
ggtaaacatt ctcagagaat atttaattag caacatgaca aaaattagtt gatttatttg 14280
aaggatattt aagtgaattt agagatgaga tttgttttta cttttttaat ttcagagagt 14340
tatgttggtt gcaaaaatct ttgttttcta gtatctattt tataagccat ctgagaccag 14400
gatgttttct ttttaacatg taaacttgat gcagtctctt tgtaaagtaa tgaatccccc 14460
cgtgaacctc aaggagaaga aagtttagct tgaacactta atttcaagta ccttgagaag 14520
ggaacaggct gtattgctca gagaagaagg ctctattagc taaggtagaa ggacaatgta 14580
aaactcttgt gaagacacct aaatgtcatt ccactcaaag cccattatta ataatttgaa 14640
actgtctgct ctgtgaaggc aacgacctat ctttactcat ttttgactct gctaacattt 14700
ctttttatag ttgaactaga ggcaaggcaa ggaacttgac atgcttgttt tccctttaga 14760
ataaagtggc cagaagcagt ttcttctgtg tgatccaaca ctttcttatc cccagagcaa 14820
ctctgcatag tctagatgta ccagagccca gcagcctgtg aggtgatgtg atgtgatgtg 14880
atgtgaacag aaaggctcca tgttggatga tgcctatgaa tctaatcaga ttctctttct 14940
tggtcatttt gatttccaga gaaggcagta gaagcaggtt ctgtgtgtat aaccagagac 15000
tagagggagg aactgacagg gaagctgggt catgagagtg gtgtgctcag cagagtaggg 15060
agtaagagag agctggctcc agagaggatg acaaaatgcc catgtctcca gcacttcttt 15120
agcattctga gttcccatcc tatcatgaat aggaagagtt ttctagctct ccatgaagcc 15180
gaaataggtt tcagactttc ctttctttcc cacacacata ataaataccc atcaggtaac 15240
ctgggcatgg ccttttattt aggcactaac aattctaaaa ggaattaata aatgcttcac 15300
tgccatttca cagtgtaaca gcatgccagt ttaccatacc attaacattt agcctcgtgg 15360
tatttctgtg ttttttcagt cagtaatgat ggcatttttg ccaagataat atagtgttaa 15420
actgtattgg ttgaaatcct aggcttttta aatgtaaact agcactttac aaagtattac 15480
aaaggaaacg aaccagggaa acttgagaag gaactttctt atccacattg ttataggtta 15540
taaaatagtt cctatttaaa ttaaaatacc tactgttgca cttttatgtg tgtttttata 15600
tataaataac atctgggttg gcaaatatgt gattttcgtc aaataggctt tgattgaaga 15660
tgaaatattt ttgtaaaact agtccttttt taaagttaaa attttatgtt accacatgaa 15720
ttgatcagca gaaatttatt tacattatgt aaattgattt ttaagagtat ccacaaggtt 15780
ttagctatta tttttgaact tgcttcctag cccacacaat taagtaataa gagtaagaaa 15840
aaagaaagca cacatccaaa tcatctatca aaacccagct tctacccaat gacacatcat 15900
tttatttctt gatcttgtaa tgttgagcac atgccttgga catattccta tatgtgggct 15960
ctattctctg cagcacgtca ctccctcttt tatccgcccc tcttatgtgc tcccactcct 16020
atcctcttca attaaaaatg cctctttctt ttttttttaa gttaattaat taattaattt 16080
ttttttattg atcattcttg ggtgtttctc gcagaggggg atttggcagg gtcataggat 16140
aatagtggag ggaaggtcag cagataaaca agtgaacaaa ggtctctggt tttcctaggc 16200
agaggaccct gcgggcttcc acagtatttg tgtccctggg tacttgagat tagggagtgg 16260
tgatgactct taacgagtct gctgccttca agcatctgtt taacaaagca catcttgcat 16320
ggcccttaat ccatttaacc ctgagtggac acagcacatg tttcagagag cacagggttg 16380
ggggtaaggt catagatcaa cagcatccca aggcacagga atttttctta gtacagaaca 16440
aaatgaagtc tcccatgtct acttctttct acacagacac agcaacaatc tgatttctct 16500
atcctttccc cacctttccc cctttcctat tccacaaaaa ccgccatcgt catcatggcc 16560
cgttctcaat gagctgttgg gtacacctcc cagacggggt ggtggccggg cagaggggct 16620
cctcacttcc cagaaggggc ggccgggcag aggtgccccc cacctcccgg atggggcggc 16680
ggctgggtgg aggcgggccc ccacctccct cccagacggg gcggctggcc gggcgggggc 16740
tgacccccca cctgcctctg ggacggggcg gctggccggg cgggggctga ccccccacct 16800
gcctccggga cagggtggct gctgggcaga ggggctcctc acttctcaga cggggcagct 16860
gccgggcgga ggggctcctc acttctcaga cggggtggcc gggcagagac gctcctcacc 16920
tcccagacgg ggtcgcggct gggcagaggc gctcctcaca tcccagacgg ggcggcgggg 16980
cagaggcgct tcccgcatct cagacgatgg gcggccgggc agagacgctc ctcacttcct 17040
agacgtgatg gtggccggga agaggcgctc ctcacttccc agactgggca gccaggcaga 17100
ggggctcctc acatcccaga cgatgggcgg ccaggcagag acgctcctca cttcccagac 17160
ggggtggcgg ccaggcagag gctgcaatct cggcactttg ggaggccaag gcaggctgct 17220
gggaggtgga ggttgtagct agccgagatc acgccactgc actccagcct gggcaacatt 17280
gagcactgag tgaaccagac tccgtctgca atcccggcac ctcgggaggc cgaggctggc 17340
ggatcactcg cggttaggag ctggagacca gcccggccaa cacagcgaaa ccctgtctcc 17400
accaaaaaaa tacgaaaacc agtcaggcgt ggcggcgcgc acctgcaatc ccaggcactc 17460
ggcaggctga ggcaggagaa tcaggcaggg aggttgcagt gagccgcgat ggcagcagta 17520
cagtccagct ttggctcggc atcagaggga taccgtggaa agagagggag agggagactg 17580
tggggagagg gagagggaga gaaaaatgcc tctttcaata tgtaaggtaa gattgataga 17640
tgattgcctt ccgtagttcc tcccaaaatt cataggttga aatcttagcc ctcagagtgg 17700
tggtattagg aagtatggag cctttgggag ataattagct caggagggtg gagccctccc 17760
gaataaattt aatgccttta taagcaagac cccagagagc cctttcaccc ttttctaccc 17820
tatgaggaca cagcgagaag acagctgtct gtgaaccagg aagcaggccc tcactacata 17880
ccgaatctac aagcaacttg atctcagact tcccagtctc ctgaactgcg agatataaat 17940
gtttgctgtt taagtcaccc agtctaaggt atttttgtta tagcagcctg aatggactca 18000
ggcggtaggc aagaggagga cagtgcacct gagttataca ttgggcaagt catttacctt 18060
cattggagcc tcggtttact tgtgttagat atgggaataa tgatagttac actgcagggt 18120
ggttgtgaag attagagaaa gtgcatatac tctgatcagc acagtgcatg gtacttcagt 18180
acacagtgtt aactgaagac acagtgttaa ctgaagacac agttgaaaac taaagatagc 18240
tagtgaaaca catgttctct gcacatcata cagtttcttg ggcagttttc aaagtgtgac 18300
tctgcagttc ataccaccat tatcacttag tctctaacaa ataatcatat ttgacacatt 18360
tggctgggac ttaataattt tatttgggca gcagctggtt tctggaagtg ttcatgggaa 18420
gaaaatgtat attaataaag ttagcaggta atgttctttg ttgaaatgtt ggatattgtg 18480
ggacacaaaa gagtcagggt aaatgtacat ttacagaatt gagcgtcaga gtgtcatagt 18540
tctctaatga tttacaaaga aagaattcct ggaacaagtg aaatacttaa taattaattc 18600
agcctgtatt tctgcaggta atcaactgca gtgataaaaa aataaaaatg gaactgtctt 18660
ttgctcattg gaaagatatt tgctgcattc aaagtcatca aaaaggcgaa gcaatgaaac 18720
ttaagacttt tgggttttaa taattaaaaa tccatcttaa agagaacctt gggctctggc 18780
acggtggctc attcttgtaa tcctagcact ttcggaggcc caggtgggcg gatcacttga 18840
ggtcaggagt ttgagaccag cctggttaac atggtgaaac cccatctcta ctaaaaatac 18900
aaaaattagc tgggtgtggt ggtgtgcgcc tgtaatccca gctacttggg aggctgaggc 18960
acgagaatca cttgaacctg ggagatggag gttgcagtga gccaagattg tgccactgca 19020
ttccagcctg ggtgacacag tgagactctg tcttgaaaaa aaaaaaaaga gagagagacc 19080
gtttgtgaag gtgtgttaca cagagaattc ctgattccta atctccagaa tagaaaaatt 19140
caaaggctca gaatcatccc aaatcaatta tccagtggca tctgcatttt ccgatttcat 19200
tctcactagc cgtgtgaaca tgggcatgac cccaggagcc aaggaattct ctaatgccta 19260
aggacagcca tgaggtcaat gaataacatg tgtcaggttt ccctggcaca gaggagcttc 19320
ccagtaaatg ttattttcct tagctatttc ccatttaaca tgtcaattat tcttagtgag 19380
tttccattca atatgagagt ttatataatt tgtcatattg aataggaact tgctaaggaa 19440
agctgtgtag cagaggaagc agccccaagt gacaggacat aaactttaaa ttcacaagat 19500
gcttgtatgt gaattatgat ggtatctgaa ttaacagtga tatttacagt ttagtaatac 19560
atttttcttc ccttgtcttg ggctttataa aaaccctgta agtcatgtat gtcaggaatt 19620
ttatcctcat aattgagaga aaaatactcc atattttatt ggaaataaga tcagtagatt 19680
ctctgtatat catacaatgt cttgcctact taaattatca ccccccttcc tatttcaaca 19740
aagactttct ctaagtaccc ctggcctctt gccctgatcc atactgttct ccttggtaat 19800
ggagtgagtt tcctgcctgg tgggtgcagc cattggccac atttctctag tgtatggaac 19860
tcaccggata aaaggcctca gatgtaggtc tctttctgct gtcttcaccc aggtgttgtg 19920
gagtgtgtct gtccttgagc actagctttc ccctgtgtgg gctgcctcaa ggcctgcatt 19980
catcaggtag ggactgttag ctggtagacc taagccacca ctccagtact gactgtcagt 20040
aaggaaggtg acatgctgac ctccatctgc tacatcattg tgtatatgtc ttcattagtt 20100
ctgcatattg gtgagaaaat ggatgctctg attcctcttg aaccccaaga gaatgaaggt 20160
aaaaaaattg aggtcctaat gcaaattttc ttagtacacc caaatctttg gatagagtcc 20220
tgcactcttt gagatatatc acacaatctg tctcactttc ttttcgcata tgttatatat 20280
tttaaaactt ttgtttttgg ggccaccact tactggctct gtgattttag gaaagctttt 20340
cagaccctca gaaccccagt ctcttcatct ctaaagtaat aaaatatatg taaagaacct 20400
atttcagcat gtgaattctg ttttcctttt tcctctagat gtatcatatc cagttaaaaa 20460
aaaatctact tgaactttgc aggtatcttc agtgtttatt acttctcatt tttaacattt 20520
attggaaatt agcatattgt cagaattgat ataatacaat atgtaacctc tttcattaag 20580
cttatcattt ttaaaataca aataactatt ctttacttaa atttttattt ctagacacag 20640
ggtcctgctc tctcgaaccc aggctggagt gcagtggcac aatcatagct cactgcagcc 20700
tcaaactctt gggcttaagt gatcctcctg cctcacactc tggagtagct gggattatag 20760
acctgagcca tggtgtccag catgacttaa attttgaata agttgcactt actaattaca 20820
cttacactgc tgcatctgaa agagtaatac aacaatagaa gccttttata gtaattattt 20880
gtggcaggta gactcctaaa atgactccca aatgacccac acccttgtac aatcaatacc 20940
ctgtcccttg agtgacagtg acttgctcta atcagttggc tttcagttaa tcaaaagaga 21000
gattatcttg ggctgaacta aactaatccc agcaagataa cagggacttc agtcttgcaa 21060
acagatatca tataaaggga atcatatctg cacatttgat tcttggaccc tgggtaggaa 21120
tttgttagag agcaaggatg tgaataaagg tgggaaataa aggtgtatct ggtgtctctg 21180
ctcaaaagct cacatgtagc tacaaggcag agatttaggg gagtggtatc cacagtcaag 21240
aagaattaat acatagtgtt tgagtctggc ttcttccaat tagcataatg cttttgaggt 21300
tcatccatat tgttgcatta ccagtagttt ttatttattt attttatcag tagtttctaa 21360
attgttgagt agtatattac attgtgtaga tatatcctgt gtgtttatcc atacacattt 21420
atggatattt gggagttgct agtttttagc tattctatga gcattcacat acatgtcttt 21480
gtgtggaaat aggttttcat atctcttggg taaatatacg ggagtggaat tgctagatca 21540
tatggtaaat actctgtata cttaaaatga agagaaactg tcaaactatt ttccataatg 21600
gctctaccat acttgatgtt ttagaaaaga ggaaaaacac tagcaatttt gatgctgtat 21660
gtctacaacc taagagaatc taataatagc gtcctggtct gtcagtgtag gaattgccta 21720
ggtaatacat ggaaggtgtg gagggagcag gtacacagaa aatgtgcgtc agaaagtttt 21780
gcttttccta tcccacgtca tttctgagtc tgatcctccc tactcctcac aaataaattg 21840
gtagattcat ctgtttttgg acccttttct gctctagtta ctgaactatc agagacttca 21900
ggaagagctt aagctaagct attccaggga agttttggtg ttactctgta cttacggact 21960
cctgcgtgtc tgtctctgtc accccagctg ccaactcaac ttagagttta ttagcctggc 22020
attgaccagc acactgtgat attctctgag gcacactgca tgctgctagt gtgccctggt 22080
tcataaagct gggccttcag cattgatccc actccaagtt ccttgctctg tcttcccaca 22140
gctgtcccct tgggggatgt ggggggtctg acatcatttg cattattagt ctattccaag 22200
aaatgcacct gtgcactaat cagttcactt tttctgtctt agtcggcaga tctagccata 22260
gcccaatccc tcaatttcta tactaagcct tttatattag taaaactgtt atgttatcta 22320
gaaattcatc agtgggaaaa ctacttcaag ggaggtagtt agaggctgca aggtgctaaa 22380
tatttttctt tctttctttt taaatgtaac ttggatctaa agtaaggacg aatttccttt 22440
tattatttgt tcagaatata ccagctggtc ctgtgggcag aagtttgtta aggggcctgg 22500
atgtgaaata acagtgtatc tcgtgtctct gctcaagagc tcacatgtag ctacaaggca 22560
gagatttagg ggagtggcat atacggccaa ctgagaaata acaatattgt cagggactcc 22620
aaggcacttt ggaagatata aagaatttct taaattttct tgtagaattg ttgcaaaagg 22680
cagacatgct taaatgttta aataagcttc ttgaaaactc atggaacatt caatcacaac 22740
tccaacagtt ctccaaaatt tgatttattc tggccatatt atgcagcaaa gaaacacttg 22800
gctatttatc taaatctctt ctgtcttgta aatgatccta gaaaataatc tagaaataca 22860
ttttattaaa gtaatgcatg aggtacatat cgattaccag ccaatagttg aacttaaaca 22920
taatgcatgg gtgtgagggc tggcattgat gattcacaat gaacacagtt gaatatcact 22980
tccctgctga gactcgggta tcatcgcaaa accacagtga gcatcagtgg ggatctgcat 23040
gctctggtca agacagatct ccatgctgag tgtctgtttg ccatgatgaa gagaacagtc 23100
aacaggagtg tttgccatga tgtagagaac agtccgggag gagtgaatgg aggatggggg 23160
catgaggaaa gatgaggctt tctctagtta tgaccctttg tgtagaagtt aggagtaggg 23220
gataaatgtt tctgcctaat atttaacttg gaagagaaga tgtccatgtg aaaaactgct 23280
aaatactgaa acaagagagc atgtgactaa gcaataccat gtgtggcaca gaaaacaagt 23340
gcaatagcag tttagcaaag tgagttcagt gtaggctgaa ctggttagag caggtttccg 23400
ggtgaaggtg aggattggtg agggtttgga ttggtagaaa gagacgggga ggacatctca 23460
catcaagaag ttatgccagt ggatttcagg agaattgagt gtttgtgtct gaaaagcctg 23520
taagaatagg gcatgacagt gtggagaaat gcatgaaagt atgaaggatc gtgatatcca 23580
gtttgaggag tgctaactag aggctctgaa gatttgagaa gaaacataat gaaaggtgtt 23640
ctcagcctga tcttggtatg caggactggt aaaccccaaa caccttacca ttctattacc 23700
tctccctcaa ggaatgggtc ccttgataag aatttagtgt aaaaaagatt gtcaattcag 23760
tgctgctgct gagtgctagt ttctttaaac acacacagtt ttcttttgag aatttttttt 23820
ctattagata gacgaactgt tatttaaatg aaaaaggcac atagtcccat aaaacaatta 23880
cacattcggg tgataacttc aaaaggagaa attaaaatgt tcttatgttt tgagcaagca 23940
tttccacttc cagactttgc tgcataaaca tctgtggtca tctagggaat gcctgacctg 24000
gttcagaggt gtcagagcaa tgtaaagtca cggaagtgct gcagttctat tctgggctct 24060
catctttttg cagcggtcca ttctctaatt ttcaaccaca tattaccaga caatctctta 24120
agtcatacac aacaaactga ttctgtttca atgcttagaa ttagaataaa aaagcctaag 24180
caaaaatagc acaaacattt gaaaaacact ccttttttct accactccct tcttagaaca 24240
gaaataaaag ccctgtactt taagaaaatg gatggaagaa ttttctttgt acttcttatt 24300
ctccaagtta catttactac ctgatagtgt taataccttt ttgtagtacc tttctttaaa 24360
atatacaggg aaatgatctg ttccaagaaa ctgtgttttt aaatttaatt atagtgtgct 24420
atgatgattt aaaaaatggc cttttgagta aaggacacga acaagttatt taaaaagtga 24480
gcagtaagtg tggtcagtaa acacatcaaa aagttccctt cactaatatt caaagaaatg 24540
caaattagaa taataatacc cttgttgctt gtcaaattaa tactttaaca attattatgt 24600
aaattttatg gataggattg caaatcaaca cacctttcct ggccactggt gtacataata 24660
gaaattctaa aaatgttctt aggcttgacc aagcatttcc acttctagac tttgctgcac 24720
aaagatctgt ggtcatctag gagaaggctt gacctggttc agaggtatca gagcaatgca 24780
aagtcatgaa aatgctgcaa ttctatcctg gactgtcatc ttttttgtag tggtccattc 24840
tctaattttt caatgacgta gtaccagaca gtctcttaag tcaaatgtat ttgtagggct 24900
ctggatttga aattcaatct ggctttcagc cttgatgtgg ccactcccaa cttgtaaagg 24960
acctgtgaag acttagacac agaataaaac acagaagata agaaaaccaa ctttgaaaga 25020
aaatttattg tcaaaattgt actgggtcaa ttgagcaaaa acttcaaaga gcgttttcat 25080
attaaccaaa gcatccctcc tcaattgaaa catataaaga tattgttcac ccctatcctg 25140
actcagagat ccacaaatct gggcattcat catcaccata ttctgctggc aatagtttta 25200
agcagctaaa ttactgttct ctactcttga gtttcctcca tctcttcaga tacagaaagt 25260
tctagaatat attcaaataa tcaaatcttt aattcattcc atatatctgg ctcaagacag 25320
tgtcccaata accttattct agagaaagtc agatttagac ttgtgctcat catcatactc 25380
atgttcttga catcatttta agctctgagg agacatctca cataactctc attgatacgt 25440
gtatacatct gtcactctgt gtttgtgctg tgatggatgg acttggatgt agatatgcat 25500
agatgatgaa gcagatagct atatatatgg tattgatgta gattcagtac agctgtataa 25560
ttgcatctgt gtttcacata tcttgtgtac tgtatcttga ctgtatctgt gtcaatacct 25620
aattgagtta taaggcataa atatgtggag ctcttagcac aatacctggc acactgcaat 25680
cagtcgatgt atgtgtttgt gtgtgtgcgt gtgtgtgtgt gtgatacaga cagaacctgc 25740
taatatgcat aatagtcagg ttgggttgtt ataacaaaat actatagact gtgtggctta 25800
aacggcagac atttgtttct cacagttctg gagtctggga aatccaaggt caagattagt 25860
ttctggtgag agcagtcttc ctcttttgca gctccccatg gtttgcaaat cacagatttc 25920
tcgctgtatt ctcacatagt gtaggaggaa cagagggagg gaagggagga agtgcctaag 25980
gacactaatc ccatgatgag agttctatcc accatgacct aattacctcc taatgccacc 26040
atctccaaat actatcacac tgggaattag gtctttaaca tgtgaatcat ttggggtgga 26100
gaagacaaac attcagctca taacaggtaa ggtagaaaat ctcaagaatt tatattttgt 26160
atatgaaaga tcattaatga gcagaatttt aagtttaatg ccaattttaa tctattttta 26220
tcttttctac gattcttttt tactttttaa tttttaaact ttcatataca aagggataat 26280
atttggctaa atgttctctt ctttttgagc tctggatcat tgttacgatc accccataat 26340
tgtatgtgtg cagagatagt gctttataca tttttaaaac aattttatat acagtattaa 26400
attttatttt tcaataaaca atccatgaga tagaaagaac atatattttg ttttacagat 26460
aagctccagg gagagtctag ggtaccttag attgtatggg taaaacatgg tttatttaaa 26520
tcataaataa acagtcttat ttccctaaat gcacatacag gcatccacat caaatgaaat 26580
cacatcacat gaattgaaca ggcagttact gacttagaac tttgcactca caaaagacac 26640
actctcctaa atgtctccat catactatac tttttgtccc caaatgccta atcactgaag 26700
cttcagactt tgttgctttg attcccttag gaaaaattcc tggtgtttca gaaaaaaaga 26760
gccatttaac tacattagaa gttaaccctc ctttaaaaat gtg 26803
<210> 20
<211> 26803
<212> DNA
<213> Homo sapiens
<400> 20
cagttaaggt ggggcaggaa taaatcacaa tggtggaatg tcatcagtta aggcaggaac 60
tggccatttt cacttctttt gtgattcttc acttgcttca ggccatctgg atgtatatgt 120
gcaggtcaca gggcatacat tggcttagct tgggctcaga ggcctgacaa ggcacagaag 180
gagctgggat agaggtgcaa agtactcatg ctgacctgac atcccatgcc cacggcagac 240
atgactactt gatcggacaa tactttcttg gaatcaaaga cacaacttca cagtcctcag 300
catcatagtt caggcagcct gtatcagtag atcagagtgg gcacatggcc tggtgctatc 360
actccatcct gtgaggggag atgggattgg aatggaggtg gaggagtcac aggtggtcct 420
gatatcacag gcctgggagt ctgaaagaag tgcagtaggt agtattatgg ccctcaaaga 480
catctgtgtc ctactgtcta gagtcaatta aagtgttacc taacatagaa aaaaggattt 540
tgcaggtggg attaaactaa ggatctcaag atagaaagac tctcctggag tatccagttg 600
tgtccaatgt aagaacaagc gttctcaaaa tagggacaca ggagattgaa ttgcaaagga 660
gatgtgctga taaacggaga ggttggagtg atgccgccat gaggcaagga atgtgggtgg 720
cctctagcag ctagcaatga ttcggaaatg gattctcccc cagagtccag aaagatcaca 780
gcagtgctga caccttgatt tgagcccagt gtcacctctg tcagacttct gccctataga 840
actgtaagag gataaatttg tgttttaagg cacaaagttt atggtaattt gttatagcag 900
caatgggaaa ctaatacaag aagggttgtg ttatcaacag aaatacagca atcagaagaa 960
agatggttgg ggagagcaga gggtggggtg ggaaggtgat ggattctgtt tgggacattc 1020
tgggtgtcat atgaatatta ttggggaact gtccatcaaa agctcagact tcaaagaaag 1080
aaaggaagga gcaacaatat tttgaaaggg agaaagtagg taatattcaa tatgtgttga 1140
gtacctacaa ggtttttagt tctttcacca agttatttta tttaattttt atggcatcaa 1200
ggggtgtttt ttttttttct taaaccaaat tttctcagtt aagcatccca tgtaagtggc 1260
caggggttaa gagatgtgac taagactaga cagagtgatt ttcatcatta catggtacta 1320
tggttctact gaagcactga ggaaggaagt caagagagtt cctgagaaat gtctggagtc 1380
ttagtaacat gcaaagtgag gcagaggtat tgtcagtgtt agctggtcac tttgaaaggt 1440
tagaggtaaa taaagttcag ggcaatggta cttggagagg agtgaatctt tggtaacctg 1500
gaggagatgc ttctcagagg aaaatttctt cacagcatga agtcaccagc tctaagacta 1560
gtgggtgttc tgggggatgt tagcagtgac tcactgactg ttggcccagt gaggggaggg 1620
atgaaaggct gggtagtggg tagagaatct gactcctcct gcccccacat gtctattcct 1680
gggccttctc ttggtggggg caaaaggtat gcaaagggat ttccgcagtg tctgagtgct 1740
cccaggagac ggaaaagtaa ctgatgactt ggagttggag tcttgttggc tcaggcagct 1800
ggggcgtttt gggatttcaa cacctccctt tagtttctat gtatggtgct gagcaagtag 1860
tcaaagactt tggtcttgct ttaccccagt agcccacaag cctgagtaca tggctaacat 1920
ttattgggca cttagtgtgt gccaaagagc tttatgagac aggttctttt tgagtcccat 1980
tatactgata atgggactga taagaaacag aaggtcagaa aggcccagta acttgggcaa 2040
ggtcatgtag ctagtaagtg gtccagctga aatttaaaac caggtttatc caactccaaa 2100
tcccacatct gatttatttt gccatgaatt aaaacatagt tctgattttg agaaactccc 2160
agtcccagtg ggaaaacaga tggaatcaca aattaccata acacagtgta gtgatgtaga 2220
tataaaaaca aagacatggg aaccctgaaa ggaagttgta acaccaatag aggaatgcat 2280
ggaggagatc tttcagagga ggtagcctcg tagctgggct caggggtgga tgagtttctg 2340
gctagaagcg cattgtgggt agagggagaa catatataga aaggcatgaa gatggtggac 2400
atgtttggaa agagtgagta ctagactgat gaacacgatg agtcaggagc ttatgacgtc 2460
aggggctact gctagcctag ctaatgactg aagattctgt gaaaagaaaa ccctaacatt 2520
tccagctgag tactagggcc atggtgatta tatatcatct ctcatgacaa cacaaatgtt 2580
tgggcttact tatcatcctg ctcagtaatc attatgtcac tcattaaaga tgtatcaggg 2640
taaattttag gtgagaattg attagaaatg taaactctat atatgtagtt acaggcttca 2700
cagtttgcca ctcaaacaat atggtcatcc aatcatgagg cacaaccttc tgctcagaac 2760
ttttttataa ttgggagaaa gtgacatccg agttccatct cttctgagaa ctgtgcagct 2820
cagaaatctt cttccctttt ttgatccccc agttgggtga gatgacatta acagtgtgtt 2880
ataagcactt ttcatgttta ctaccccata ttcaaaatta agcatttcaa acaactgtat 2940
aattgtctat aaatgtagca ttattagtta attttccttt taacacattg aggttacctc 3000
taatgttttg ctatttttaa ctatgttgaa ataatcatgt ggtttttatt ctttgaatta 3060
ttttatattg taatgttccc tgaaatgaaa ttagttgatc aaaagatgag taatcttttc 3120
atgacttttg atacatgtca ctcaatcacc ttctggaaga gttggaacaa gagcctattg 3180
ctggcatccc atttcatagc catctcacca atgttggaaa atgggctttt cctattttct 3240
gctaattcat tagttgttaa tggcttattt taactattaa tcccttgtta ttgatgaaag 3300
ttaagcattt tctgtatttt tattttctgt ctaaattcta ctttttgcac aatttccttt 3360
gctcacttct tcactggaat ttagtatttt tctttccaat ttgtatgact ccaaaatcag 3420
tgataaaata agcaaagggt ctttcttaga ggctaaacaa gggaatgaat aaggaacaag 3480
aacagcaaag ggatagtgaa taaagataca aaataataac ttatttttat ttttgaagct 3540
ttccccaggc agtaattctt gaccttttta agattataaa ttattttgag atatgcattc 3600
ttatcctcaa aatgaattag tacataaaat ttaaatgggt tgatagatcc cctgaagtgt 3660
atccacaaaa tgctctcagg ttaagcaaca ttgttctata ataactgctt taaccaaaat 3720
gtttaattat tgcttgtgtt tacctgtcct agcaaagagc taattgttaa attttgtgtt 3780
agagggtcaa tattataaat aacttaatgt ggcctctctt tcctttcctt tttttgagag 3840
ttttgaggct atcactaagt gggctttaga gtaccaggtg ggaatctgac ctcactgctc 3900
aaactttcaa cttcaggcat gaagtggtcc cagtgatgtg ggaacctcag agtctaaaac 3960
aaaaatagta gactgaggcc ctcagaaacc agacttatca gattcagaat ttaatataac 4020
tatgtttgaa agatttaaag aaaataaatc aaattaaaaa gtgagcaaga agcaagaata 4080
acaaatgaac tggcatattt gagaaagaat tttgagcttt tagaaataat acatacactt 4140
attgaaataa aaactcaagt caataggttt aaacagcaaa atgaatatag ttaaagaaaa 4200
ctattgtgct gaaagattta aaaaactatt acacagaatg caaaatgatg aaatagggta 4260
atttaatata tggcagatat gttaagacac tatgaggata gtatgagaag acctgataga 4320
catctaatca cagcacctta aggtgaaaat agagagagtg caccaatatt gaggagttaa 4380
tgtctgagac tattccagaa ctgataaata gatgaatcta cagatctcag aaacacagct 4440
tttacaaagg ataaatgaag agaaattcac cagagataca ttgtaatgaa tctgtaaatc 4500
accaaagcta aagatattat ttcacaagtg gaatgacagc tgacttctca acaacaacga 4560
aagcaaggag acagttgaaa gacatcttga aaatggtgag agaaaaacta actgttaata 4620
taaaattgtg tacctatcaa aaatatcttt tggccaggcg tggtggctca cgcctataat 4680
cctagcactt tgggaagcca aggaacgtgg atcacttgag gtcaggagtt tgagaccgag 4740
accgtcctgg ccaacatggt gaaaccccat ctctactaaa aatacaaaaa aaaaaaaaaa 4800
atagccatgt gtggtggtgt gcacctgtaa tcccagctac ttgggaggct gaggcaggag 4860
aatcgcttga acccaggagg tagaggttgc agtgagctga gatcatgctg ctgcactcca 4920
gtctgggtga cagagtgaga ctgcctcaaa accaaaccaa accaaaccaa accaatcttt 4980
caagaagtac accataaaga catttccgga taaacagtga atgagattac ttcctaaacg 5040
aatatgtaag gatgtttctt tttaaaattt tttttgggtg tattcatcac ctcaagcatt 5100
tattcttttg tgttgttaca actgataaga atatctaagg atgtttcaaa aggaagaaaa 5160
atgatcccta aagtctggta tattagtttc ttattagttg ctctaagaaa ttactgcaaa 5220
cttagtggct ttaaaaaaca cgaaattatc tttcagttct gtaggtcaga aatttaacct 5280
cctgatttct ggtcagaagt cttactgggc taaaatcagg gtatcagcag ggctgctgga 5340
ggctctggag atgatgtttc tctaatttct agaggcattt gcattcctta gctagtggct 5400
tcttcccaca tcttcaaagc taacagtggc agtcgagtcc ttctcttatc tttttttttt 5460
ggggggggtt ataaattttt tttattatta ttatacttta agttttaggg tacatgtgca 5520
caacgtgcag gtttgttaca tatgtataca tgtgccacat tggtgtgctg cacccattaa 5580
ctcatcattt agcattaggt atatctccta atgctatccg tcccccctcc cccaccccac 5640
aacagtcccc agtgtgtgat gttccccttc ccttctctta tcttatcact ctgacctctt 5700
ctgcctcctt tctctacttt taaggaccct tgtgattaga ttgggtccac ctaaataatc 5760
caggataatc tctttatttt aaggttagct gattagcaac ctcaattcca tctgtaacct 5820
taatttcttt tttgccatgt aacctaacat agtcacaggc ccaagggaat aataggatgt 5880
gggaattgcg ggtggtggag taggggcatt attctgctta ccacagggga ccttggatgt 5940
cagtaaaaaa tggtgaggaa gaaaacagga aacgtagaat aatctaaaca aatctttgaa 6000
aaataagttg aaatgtgggg ttagaaaaag ggcagaacta aaatgttaag acaaattaaa 6060
atgttaagcc tataagttga gagggagatg atcaaggtta aaaatactca taaatcatat 6120
ttttttgggt agggggttag atatttatta attttagacc ctgagaagta aaatagtcat 6180
gacaaaaatt taatagtaac cactaaaaca agagaaacag agtatataac ttctaaatca 6240
atatgggaag gggaatggaa taagaaaaca atagcaacca ccaacacccc acccaaaata 6300
aataataagg tgagagagaa aaagaagcat tgaaaagggg ctaaaataaa ataaaaaatt 6360
aagtcatgcc cgtaatccca gcactttgag aggccaagat gggtggatca cctgaggtca 6420
ggagttcgag accagcctga ccaacatggg gaaaccccat ctctactaaa aattcaaaat 6480
tagctgggtg tgggggtgca tgcctgtaat cccagctact ctggaggctg aggcaggaga 6540
atcgcttgaa cccaggaggc agaggttgtg gtgagcagag attgtgccat tgcattccag 6600
cctgggcagc aagagcgaaa cgccatctaa aaaaaaaaaa aaaaaaatta agatagtaaa 6660
acaaatccaa atacattaaa gattacaaaa aatgaaaatg aactaaactt cctagttgaa 6720
agacatattg tcagattgga ttttaaaatc tggcaatgat atttccagga cacagtccta 6780
aaatattaaa cacagaaaag ttgaaagtaa agggatggaa gaacacagtc aaactaacta 6840
gaagagagct gggttcaccc ttttatatca gagcagaatt tcactgagaa aacactaaca 6900
ggaaggggga aaggcacaac atagtgatga agcacttaat tctcaacaaa tgtatgacac 6960
ttctaaacta ttatgactgc aaaaaaaaaa aaaacctcag catatataaa attttggaag 7020
agccatggct atcagctgac atgcacttac catatttgca ttgtcctact acaaacttta 7080
cccatatgac caaggtgctg tcttcatccc cattttacag atggggaaat ttccccatag 7140
agaaaaagat ttgggccatg ccttggtttc ttcttgactc tgcttcctgc tgattgatcc 7200
tcacacctca cctggagacc tacttcacat tatccatcag gatgagaact ggtgtgcctt 7260
taccaaagga ccacagtcac cttcaaaacg tgtttagata gatctggatg gagacttatg 7320
tggttgcaga tgaaaaaaaa gtccccatgg tatatctgac tccatgtcat tcactgcatg 7380
aacactcaca cacatgcaca cgtacacagg caaacacaat cctgttctgg tccggcagta 7440
gtgccaacac tcaaatccca ggtcctactc tgaagccttt gaggaaacca ttgtcattct 7500
ggggtttatg ggaatatttc ttcttggtgc cccttgttcc tcacagaagc tttttttctg 7560
tgctgtgggc ttgctatcct agctttccaa gctatcctag ttttgcaaat ttctctgatc 7620
aatcacatca ggttcttgga tcatcccaaa attaaagagg tacgttttgc tgcaagagac 7680
aaagacattc acctctcaca cagtttgaat tcctgggtag aaacagattt caatgctggc 7740
cagcctctgt agctcccagt gccccctcaa ttatgactcc cgaggcattg gagcaggatc 7800
ctcaccttgc gcaaagataa ttaggtgtgc tgaagaaagc cagacactgc tctttcttag 7860
ctctgcatcc ttaaaaagtt accactactc cttctctttg aatctttgac ttgtcaatgg 7920
taaagtgcct atgattctta aagggataaa aacgcaacct atataaagca cctggcatag 7980
tgcctggcac aggtgggtgt ttaacaaatg ttaactcctt tctatcccac tgtgggtgtc 8040
catatcctct tgggtaggct gcctctggtt tccattgtgt catcagagcc atactaaatc 8100
gatgtgttgg acatggatcc caagaactaa tttctggaat gagttatgcc taccaacttc 8160
cctgatactt acttctgtct ctccatttat caaatggaaa tgtttgcatt tggcctctgc 8220
cagttggagt aaccaaaagg cttagaggtt ttggagaggt cctacactgc catctgtttg 8280
accacatact gcctttcacg tgtaataagc attgtttctt acatttgtct aggaattagc 8340
agttcacaaa gcacattcgc atataagggc ttgttttgaa ttgatcttgg cagcaattct 8400
atgagacaag taaaaggtag gtcaagcatt ataatcctca ttttaaatct gggaaaactg 8460
aatctcaaaa aggttgaaag acttgtctag gggacagtgt gtgggtaaat gagaagttaa 8520
gatttgctga actggcattt cctgactaca tatctagtgt ttatttatgg agaaggcact 8580
acggtggcca agtggctcag agtcacacag ctctgtgcct cagtttattt gtctgtaaaa 8640
tgaagataat aatatattct gaatactgtt gtggaatttc attgagatag ttcacataat 8700
ggcatggata ctgtagtaca ccgcctagat tcacaacccc acgactatcc atgagaatgg 8760
cccatagctc aagaggtcac attctttccc caggtgaagc ccgcatccaa tgactgctca 8820
gtgtgggtat ataaaggcct ggctcccttg ccagtgccac cctacctcct ggcgaatgga 8880
ctgaggcttg cattatgact gcattgcagc tcaactgttc tttccattca gtctttcctt 8940
tacactcaca caggtgagga cccagccatt aaatgcctca catgatgacc tctattgcat 9000
gctaggcttc gtgtgcacac acaaaaacca tcctctatca gtcaggcccc ttacttgcag 9060
aggcaaagag gccagtctgg gttagatgtg caccctgttt ttacaacaga acgagacaaa 9120
caggtcctcc ttaggtgtaa gttcatggcc ttggccccac ccctgaaact cagccatctg 9180
agacagtttt aggactgagg caaccccaga ctttgggttg ttgtttgctg ggcacagcct 9240
ccttttgcca atgttgcaat cctttgtaag agaccacagg ttgatcaccc gttcctgtta 9300
ctgagcacag agaggtttgt aggccagtct ctccaggaat tctgacgcac tgcaaaatcc 9360
catggtctga atgcttctac cttccctgtg aaagcccctg gcctcagaaa aggaagctgg 9420
tttaagtgac caacatttgg ggtggagctt ccgagggccc agccaccgtg ttacatgcaa 9480
caatcaacag agatattctt tggctagagt tgtttttctg ggtataggac cctataatta 9540
aaatcagctc tccaacttcc cttcctccaa aaagtataca atgtaaggag gaaaatgcaa 9600
attgaaaagt tgctagccct tcccgccaga atgccacccc aagcctcccc tgcaggaagt 9660
tctggagtcc ccaactctgc cccaagcctg aggcccttga taaggtaaag ggatgtgagt 9720
gtggaggccg gagcccccct cgccctgtag gctgctcccc ttgcttttcc ctttgaatgt 9780
tacagtttag ttctgtgact tacttataat tgcctgaatt ccccgcccac gctttctctt 9840
actttggggc ttctcaccgt tcctcttcct ccatcttcct cctctgaccc cactcccaag 9900
cctgaatggg gtgttcctct tccgtgctcc cacaaaaccc tgtgcctctt tcatcgttgc 9960
atatattatg ctgtaatgta gttttctgga ggtgtgaaga ctatgaatcc tatgaagaca 10020
aatacacacc taccttgctg accgttgtat cctcagaatc taatactgca cttggcacac 10080
agtggttggg agtaaatatt catgaggcaa acacacagga aggactcttt tgttatggtt 10140
gtttgttgtt gttgcaagta acataacccc accttgatct agctgactga cggctctacc 10200
accttgtctt ttgcagcaca agaggaatag gaactgcacc tcttccttca gtttcagctt 10260
gaataatatc aggaagattc gtatcggtct gagttgggtc acgtacccga cgtgctatag 10320
ctgaggatgg ggtaagctga ttggagtttg caacactgtt cacatagcca agatatggaa 10380
agaacctaaa tgtcaactgg tggatgaatg gataaagaaa ttgtggtata tacatacact 10440
ggaatattat tcaaccttaa aaagaaggaa atcctaacat ttgtgacaac atggatggac 10500
ctggagggaa ttatgctgag tgaaataaga cagacacaaa aagacatttc ttgcaggagc 10560
tcacttatat gtggaatcta aaatagtcaa gcttaaagaa gagagtagac tactggttgt 10620
caggagcagg agaaaagtgg aaatgaagag gtgatagtta aagggtacaa agtttcagtt 10680
atacaagata aataagttct ggaggtttac tattaatata tcacatagta cctataagta 10740
acaatactgt attgtatact taaaattgct aagagggtat atcttatatg ttcttaccaa 10800
taataataat aatggtaata attaaggggc aggaggacac ttcaaaaggt gatggatatg 10860
tttatggcct tgatggtggt gatggtttca tgagagtata cttatcccca aactaattga 10920
gatgtaaata ttaaatatgt acagcttttt gtatgtcaat cgtacctcag taaagtagtt 10980
taaaaatggt tggactgaga aaaggaggag ctgctcagca acatgaggct gggtgctggg 11040
cagacaaaac ctcacacatg cattactgaa cccacggact catgtctgtg agctttgtgg 11100
ctgtggatgc accatgccaa tgtagtcaag gattcttcaa tgtgtacctt actggattag 11160
ttccctctgt ttatttgtat tgactcctca gttcccttat gggttgcttg ttggctcttg 11220
tggaattatt taagtaaggc tttggttgca ggagtcagac atgcttaagc tggttttggc 11280
tgtaatgggg aatttgtgag atgtgacaca gtgacaggaa gtgcagccag acgttgtgag 11340
aggcagtaac tgggaatagg aaagttatga gaagttaagg cagtagtaat tgtttctcta 11400
tcgaaggcca taatgttatc attcctgcct ctatctgttt gcttgttctt ctctctcagc 11460
agaatggtct tctctgcttc tctgtgcacc tgcagaaggt gaccacccta aggcttatgg 11520
attcaggagt cctcagtttg agagcagtta ccaaatgcct cgcccctgtg aactctagtc 11580
ccagtttctt attcctggta ctggcttagt ttgaggcagc tgcccaccgt aagtccagtg 11640
agccatggcc tggggacaag gtcaggtaca tgtgtaatca gctgggtcta tgagttgtgg 11700
tggtggcgat ggaggtagct cccagaaaaa tggattacag gttaagaaga gtctatatat 11760
cttgaccaca tttgatgtga cttggaagtt tcaatgtgtg accacttaca aagttatgat 11820
caggtgtctc atttatttat ggtgatgtat gagtccgtgt gtgagtgtgt gtgtgtctgt 11880
gtttgtgtgt gtgtgtgaga gagagagaga gaatgagaat atgagtggtg gtttcccatt 11940
tatttttcta tgggccaaat cctgggctat gagctggaga tataagagta aacaaaatag 12000
acacggtgct tgctgtgacc tgccctggga gacagatatt acacggagga tcacataaat 12060
gcagagtctg agctgaggtc ttaagggtga ggataagtaa ctagtcagtg gagcagggga 12120
gggaagggtg ttccatgcct gtgtcaaggt cctgaggtga gagaaagagg tgaatttgag 12180
gaaccaaaag gagttcaggg aacaagaggg gctgtttcac aagctgagtc tggaagctga 12240
gcaggctgag cagattctta ggaactctgc aaaaacttaa gggcttgtct atgaccgtaa 12300
acaactacgg tcgtagataa acaagttggc aaacaactca aggatttaca gtcatcagaa 12360
ttgtaaaact gatcccatgt attttcaaac ctcaaatccc ttgctctgac cttgtgtgtc 12420
taagctagaa ggaagcagga tgcacctctc ccctggctgg aatgaaggga ttcacccaag 12480
ctctcagtct tctcacggca tccagggccc ccctgcttgt gtgtggtcta gatttccatt 12540
cccatagtag gaattccttt gggagccttg gggtctctcc tgctagaggg cttcacctgt 12600
gactgtctca attcaaggga ggggttctaa taacattaaa cctcaatatc ttgctcttcc 12660
attcctgatg cctccctttc cttctaccct tcccctacct ccttctttcc tatgccacag 12720
gctggcaggt tagtgccaga gcaagtggcc agtcaccatc attggggttg tggatcctca 12780
gggttcttca gaagcccctt caccatgatc aagagtctcc agtcactcag aattccacgg 12840
ttcccaaatg ccagctctcc cactactccc agcgttctcc atctctggga tgttgggctc 12900
caggcttctc agatgcactg agtaccctag gctaagccac tgatccatca gaacttcata 12960
gcctgagagg agagggagac aggcttgcaa aggagagttc tgactagaca ctggggtgct 13020
acagatgccc gtaagttgtc cctttaccct cgatgtcccc agtgttggga ccacctggga 13080
atgcccagcc ttgtgtgctg attgacttgt agtcccctgg ccctgactgg aagcctagtt 13140
ttctctctca tgaccagcta ggcccctagc tccccaggga agaaatccaa tctatttcct 13200
ctgatagtaa tggctaatac ttaaataatg ccaaccacat gccaagcact ttacagttgt 13260
tagcccagtc ttcacaagca ccttgtgagg tgggcaagag tcttatctta ctctcatttt 13320
atagatgtgg cccaggatcc cacagctata gttcatggtg ctgggatttg aacctctggc 13380
caccagagcc caccttaatg tgtcctcctc ctgttgtcat aacagaaaag tacaacacca 13440
tgatgacaca tcaggctatc ctggcaggtt cccaggctgc cccaatgccc aactttctag 13500
gtttacaaag ttgacattta cgaagtttcc aggtttacaa atctagtttc tgattcttta 13560
gtcagcagga atttctctac aaaagctgct tcgaaaattt ccagccaaac cttacacacc 13620
ttggcattac atcttggtga gccaaggcgg aagagaacag gaagtgaagg ccccatggga 13680
agtccctgcg gtcgggagca cccaggcggg gcggggggtg gggggctttc ctgtggccgg 13740
ctccctgccc ctcccacccc cattcaggcc ctgtgagttg aatgaagaga ccctgggaat 13800
gagtccaggt ctgcagggtt agaggaaatt gaaggccctt accagatccc tgttgagaag 13860
tttatgaatt atgagccctt ctgcaaatga gagggttctt ccctgtcagg agggacagat 13920
tgtaggtggc aagattggtg gcagccagta ggctggtctg ctccttcctc tctatttcat 13980
atgtgtatga aggcattacc tgcagcaagg gcctgtgtaa atgcatgtga tttacagagc 14040
attttatgta ctgcgtgtca ttcatgcttc cggtgagccc taagtctaag atagggcaga 14100
tagcatcagg tccattttgc agctgtcaaa atgaggtctg aagggcagaa gtggtgtgcc 14160
cacacacaca caactggttg gctgcagacc tggggactag acccgggact tcgtcctgcc 14220
caggggtctc ttgccactgc tccccatcaa cttggatggc tttaagcatt tgtgagttgt 14280
ctgctccctg atggcagaat gcagagacat gaagctacaa gcaggttcgc tcccaacggc 14340
aaaaaggagg aggggtgttc agaacatcag gtgcttctag agaaagcagg gagagagtat 14400
ctggccttgt ggacaatgtc acggcagagg ccaggtatag ggcatggggg taactggaag 14460
cgggatggac cctgttattc cctaagacat ggcttccacg tagtgctcaa acaaggcctt 14520
tgcccttgct gttccctcca cctggaatat tcttcccctt ccttgacatt gctcaggtct 14580
ccactcttat gtcaccctct cagagagggc ttccctggcc actttcccta aaatagccac 14640
ccactcctag gtccctcaaa agcatatcct gctttggatt ttccctatag caatatgccc 14700
tatgaagtta ttttatttgc taacttgttt cttgtctgtt ttcctttgtt agagcgttgg 14760
ggaccttgtc tggcttgttc ccaatgcctg gaagagtgcc tggcacacag gattaagcca 14820
acacatatgt tttgaatgaa tgtgtgcaca catgcatgag ctggcggcag tcggggttgg 14880
ggtaagcacg aaggcccagc tcagttctct gcatgtgacc tcccatctta cgcagataag 14940
aaccagtttg gtttctgcta gcctgagtca ccctcctgga aactgggcct gcttggcatc 15000
aagtcagcca tcagccggcc catctcctca tgctggccaa ccctctgtga gtgtgtggga 15060
ggggaggctg ggctcctcct tgtactctct gaggtgctct ggaaggaggg gcagctccac 15120
cctgggaggg actgtggccc aggtactgcc cgggtgctac tttatgggca gcagctcagt 15180
tgagttagag tctggaagac ctcagaagac ctcctgtcct atgaggccct ccccatggct 15240
ttaggtaagc tccttccact ctcatttttt cacctgagaa atgagagagg aaaatgtcta 15300
caattggtgt ttatcaaatg ctttcaggct ctggtgagca agcgtccagg aaaatgtcaa 15360
gcgcatggag ctccaggcct gtctggggga tctgggcacg gggaggcatc catgggagac 15420
catgcaggca ctctgaggca ggggctgcaa gcctagtgcc tgctggggca gcaggtgaac 15480
agagaggtgt aactgctgtg acagaagtca tggagtcctt ggagtgtgag ggtcattttc 15540
cactgttgat agaataggga aattggtgaa atagccctgt taaatgagag aaagaacagt 15600
gtgagctcaa tgagaaatac taatagaatg tggcactgag ccacaaggtc tgaggcttga 15660
ttgataagga agggtgggga ctgtggagaa ttaagggctt ggcacaggtc agttccacca 15720
gttgtcacaa gagaatgcag gctcaggtgg ccagaacttc tcgcttttcc agaagagtcc 15780
gatattctga tttcattata tatagtattc tgattaaacc agacaataaa gcaagcagat 15840
aaaatattta aagtataagc tgccagtttg caacctccgg ttaggatttg tgtggggcaa 15900
agaaaaaaac tctcaggatc attggtatgt agactctaat tttaagtttc taatttaaaa 15960
ttggcccctg aggctgggcg tggtggctca cacctgtaat cccagcattt tgggaggcca 16020
aggtgggtgg atctcttgag gtcaagagtt caaggcctgc ctggccaaca tggtgaaacc 16080
ctgtctctat taaaaataca aaaattagct gggcatggtg gtgcatgtct gcaatcttag 16140
ctacttgggt agctaaggca ggagaattgc tggaacccgg gaggtagagg ttgcagtgaa 16200
tggagatcac accactgcac tccagtctgg gcaatagaga gagacgctct ctctaaaaaa 16260
aaatatgtaa agataaataa aatgaaataa aataggcctc taatgagcag gccattctcc 16320
tttctgggtc ttactttcct tgcactcctt tctgggtgtt aagaggaggt ctagaggaag 16380
ctggacaact cttagcttgt agtaagcaca gtggaagtat cagctcttaa tgggtcatgg 16440
acacgttaca agctaggcgc cgtgctgagc actttacatg gtttatccca ctgaaccctc 16500
tcaataaccc tatgaggaag ggctattatt gctcacattt tcagaagagg aaatggatat 16560
agagagatta gataatttgc ccatggccag acagctagta taagaggagg aggtggattg 16620
actgcagaca ttctgtcttc aaaccactac actatgctat ggaggcacag agacttaatg 16680
aaatcatgga gaggggaatt gctttgtcaa ccacaagcag ttattccggg ggcagcagat 16740
cctcccctgt cccccagtgg gtacaatggt ccctggtggg ttgtgctaca atgttagccc 16800
atggtcttat gtgtttttca aatgtgtaaa gtaggatgct ggaaccactc ttagaaccag 16860
ataccaatac attgtgaaga aataaatctc tgtgcttaaa actggttcat cccaaaatat 16920
tttgaactga cacacaatag gtgctaaata aatgtgtgtt aacttgaatt ggattgaatt 16980
cgggaaaaaa gtgcaataag cttagtgaag acaccatgtt ccctgggtag aggaaccaca 17040
ttctccatct aaggccagga gtatgggagg tatcaatgtt tgcccagcac agaacagggt 17100
gccaagaaga gaaaagttga cggggtgcat actctgactg gaaactggaa gggtgagaac 17160
agagggtaaa ggatagagat ggaaccatgt gcatacactt tgtgttacct tggacaagtc 17220
attcatttct ctggacctct gctttctctc tacacaatgg ggtcccacca cttcccttac 17280
agctgacttg tatgaagaag gaggtggagg aggaggagaa ggtgaagaca atgctgactc 17340
aaagggtaaa ttatttttag gatccaagtt tgaaaacaat tttaggctac tagatatgaa 17400
caacatcttg attatgtagt tgaaggaaat taaagatgaa tggtttaatt aaaaattaat 17460
cagaatgaaa acgattgatt actaatatat ctgcaatggt ttattttcct gagtggcaga 17520
ctcactaagg tttttgaata ctcctgtgtg attgctctat gtatgtatgt atgtatgtat 17580
gtatgcatgt atctatctat ctgttgtcta ataaaatgga tcacatctct gctaataaaa 17640
acactacact ggcagggtac aattataatc attaactgtg cctggaattt gcagcagcag 17700
ccaccagagg taccagtgcc ctttaagggt tcataattta gaataatcca attatctgag 17760
tttttcaggg actgaggggt ttggcaaggt gtagaacttt cagtaataaa gtcaagaaag 17820
tcctggacaa accaaggtag ttggtcactc tagtccataa ccaggtaaag agctttccct 17880
gtaacctgtg taaggtttta gaatcatttc tttccttatt accaaaaatc ctccccaaat 17940
tttcaagaaa ttatgaacta aatagttact ctatgagata ggagttcagc ccaaaagaaa 18000
caccataaga acaaatataa ttcttgctta tgttaaccat gcaatgaagc agagagaaaa 18060
agtcagtggc ctctttagga ggactgtagt gtgggaagaa ataactaaac tgggtttcaa 18120
tcctggcctg gccaggatct ggagcaagtg agttaatctt tctaagcctt gagtagtttc 18180
ttcttcttct tcttcttctt cctccccctt ctcctcttct tcttcctcct ccttctcctc 18240
ttcttcttct tcttcttctt cctcttcttc ttcttcctcc tcctcctcct cctcttcttc 18300
ttcttcctct tcctcttcct cttcctcttc ttcctcttcc tcttattctt cttcgtcgtc 18360
ttcgtcttct ttttattttc aaagtgaaag caagtttatt aagaaagtaa aggaataaaa 18420
gaatggccac tccatagaca gagtagcctg aaccttgagt tcttctataa agtcactatg 18480
aatttatact cattttgaaa gtgggtgtca atatgtctgt ccactttgca cagctgttat 18540
gtggacaaaa ggagatctgt gtgaaagtgt aacacagagc ctaaactata acaggtaagc 18600
aacacagttg tccccttccc catggtgtct gttcttctcc atttcctcct gtctgcaggg 18660
ggattataaa actaatcatc aaagccaaga aggcaagagc aagcatgtac cgctgaaaac 18720
acaagataac tgcataagta atgactttca gtgcagattc atagctaacc cataaactgc 18780
tggggcaaaa atcatcttgg aaggctctga acctcagaaa ggattcacag taagttaacc 18840
atgtagatct gagaggagag tagcttcttg tagataacag ttggattata taccatgtcc 18900
tgatcccctt catcatccag gagagcagag gtggtcaccc tgatagcagc aagcctgggg 18960
gctgcagctt ggtgggtaga ggtactcagg ggtacagatg tctccaaacc tgtcctgctg 19020
ccttagggag cttctaataa gttgatggat ttggttaaaa ttaacttggc tacttggcag 19080
gactgggtca gtgaggacca acaaaaagaa gacatcagat tataccctgg gggtttgtat 19140
ttcttgtgtt tctttctctt ctttgtacta aaatatttac ccatgactgg gaaagagcaa 19200
ctggagtctt tgtagcatta tcttagcaaa aatttacaaa gtttggaaaa caatattgcc 19260
catattgtgt ggtgtgtcct gtgacactca ggattcaagt gttggccgaa gccactaaat 19320
gtgagatgaa gccattacaa ggcagtgtgc acatctgtcc acccaagctg gatgccaaca 19380
tttcacaaat agtgcttgcg tgacacaaat gcagttccag gaggcccaaa tgaaaatgtt 19440
tgtactgaaa tttgttaaag cttcccgaca aactagattt atcagtaagg attgttttct 19500
gcaaggggga tgaaacttgt ggggtgagcc atttgggctg aggaggaggg aggttggagc 19560
tgagaaatgt ggagacaatt tccctttaga aggactgaat ctccctgcct ctctggggtg 19620
cggcagccag caggatccaa tggtgtatat gtctccccag ctccccattc agtgatatca 19680
tgtcagtagc ttgaaattat ccgtggtggg agtattatgt catggaaatt ggcaaatgga 19740
aacttttatt ggagattcaa ttgttaaact tttaccagca caacactgcc ctgccttcag 19800
agtcaatgac cctatccaag tttaatccat ctgtccactg tctccaacac gatctttata 19860
aaacacacct gacaacatta cccttttatt cagtttttta aaagataagt ttccagctca 19920
tcggggtggc tttaaaggcc atttctcctc tggacctcac ccaacttttc aaatcacttt 19980
tcctacccct acctctaaat gctactcaaa ctccagccat cctgaataat aagacttttg 20040
aaaagtagat tatgggctgg gcacagtggc tcacacctgt aatcccagca ctttgggagg 20100
ccaagatggg tggatcacct gaggtcggga gttcgagacc agcctgacta acatagtgaa 20160
accctgtctc tactaaaaat acaaaattag ttgggggtgg tggcacaagc ctgtaatccc 20220
agctactcag gaggttgagg caggggaatt gcttgaacct gggaggcgga ggttgcggtg 20280
agcctagatt gctccactgc actccagcct gggcaacaag agcgaaactc catctcaaaa 20340
aaataaataa ataaataaag tagattacat cagatacctc tggcctaggt tgtttatgac 20400
caactctcct gctgagaata actagaaaag ctagacaaaa catatttcca aaagatctct 20460
ttggaggcat cagagaatgg ccaaggctgt aaggaactgc ctgagcccag agaggtggag 20520
cccagcactg gtgcccttta ctcctgggga catgtgctgg tttcaaaaac ttcagctgag 20580
cttttgagca ttcatggaac ttggtggggg agatgaaatt tgtaccttaa atcctgccta 20640
cagggagggt ccctgataat ccccacccaa tttggaaatc tgggtcagcc ttcacaggta 20700
ctgaagccct cctctgaatg atctcaagtc ctgctagggt agaggttacc tgcttttgaa 20760
aggctcctgg cctacctgtg cagcaggagc aaaagtgaac catctcaggg tacagataac 20820
aatcatccag agccttgaat gacctctact gtgcttaata tatagtattc agcagtcagt 20880
aaaaaggatt taggcacatg caagatgacc tgtgtatcag ggagaaatag gcaataaatt 20940
gagatccagc agggatttga atcatggatt tgaatcaggg gcagccttcg aaagaactat 21000
ggagaatata ctcagattta aaacataaga ttggaatttt tggcagagaa ctaacaactg 21060
tacaaaaaag gaaccaaatg gaaatcctag aactgaaaga tgcaattaac cgatgttgag 21120
aaatagccaa catctattga acacttccca tgtggacagc tgtgctaaac actttacagg 21180
catcaacata agatgtgtcc ccttacagca gtgcagtgtc cctcctaaga catggacagc 21240
ctggtttccc tatctctctg cttcatcaaa acccctttac gtggggctta gacactcctg 21300
ttgtctctag tgtctagtag cacagggctc agcacatgga agccactaga tacaatttga 21360
tgaccaggac ctccgatgaa agccatgggt gctgattggg aaggcattgt cttttatgtg 21420
ctatggtctt aaagcttcat ccaggaagca gaactcgggg ggtgctgagg acccagaacc 21480
gagaataaga ttagtcagag atttcctgtg ggcagaaatc ataaggacgc caactgtttg 21540
ggtgagataa gacgaaacca agagtggact tgtggccaga agcgtgagga agagggagag 21600
agcttccctt gtcccctttc ttcctctccc taagccacag tgattgacag ccccccccct 21660
ttggagtcag agcaggcttg agactggact gggaaaggag ggtgggtcag gatacagagc 21720
aggaaggctg ggagtgcagg gcaggagcaa ggggctgggg cattcattgt gcctgatctc 21780
tcccacttta cctggggtaa agaagcatat gcaaaagcca cggtgtgagt atttcccaag 21840
tgccagggtc agggcatgat tcatcacgtg cagcatttca ttcaatcctt atagtaaccg 21900
atgatgtggc ttctattatt agctctatca gataatgaaa ctgagaccaa gacaggctct 21960
gcacattgtg tggggtaatg acacaggggg attcagacct agactccata actcctgccc 22020
cagggaccac ccccaccctc accctgtgca tgtcgacaaa ggacagactg ggccacttct 22080
caggacacag cggggaaatg acacagagca gggaggttcc aggagccccg agcgtctttt 22140
ctccaggaga atactctctg aattcagact ggggtcagag aaacatttac ccaggagccg 22200
cagtgtgggt ggggcttttt acttgaaacg ctgtctgaag gcagtggcca ggatggaact 22260
ctccacccta ccttggcaag ccacttctct tctgcaatct gtaaggacat tgttgagaga 22320
attatggtct tccaattccg gagggttgaa gaaagacaaa taggagagaa cctatcatag 22380
tcaggtgcta gctgccttct ctttcagaga gtgtgagaat aaagtgatac acttgattat 22440
tagcaaatac tttggaaatt ttaaacgcta atattcaaca cactctggaa gaggcaaata 22500
agtagacagg ttcatataca tcatctcctt cagctagtcc tcacaaaaac aaacaaatga 22560
ataaacaaaa ttcttctttg gccctcatag gaagacactg tttcttgaac gtgtttcaaa 22620
aaggatgggt gactcactca aggtcacact gtttatgagg acagtacagg aatacagaca 22680
tgccattttg cctgaaaaaa tccatcaccc agggaggtga cacaattttg cagaaatgtt 22740
ctatttcctc tgaaggatac attctttaaa cctttgggaa attcattcat agtcttcctc 22800
ctttgaagga ttaactctct ggacacaaag tgtttgattc tgatttgttg gttggaagat 22860
gtgttggttg agagaaagat tctgatttgt tggttgaaaa tagactcatc aagatcaact 22920
gctgtagtag taaatatttt gacattttgt ctgtattcct gtgctgccct cacaagctgc 22980
atcaccttga gtgagtcatt catacttttt tgtttgtttt tgttttggag atggagtctt 23040
actctgttgc ctaggctgga gtgcggtggc gtgatcttgg ctcactgcga cctccatctc 23100
ctgggttcaa gtgatcctcc tgcctcagcc tcccgagtag ctgggattac aggcacatgc 23160
caccatccct gctaattttt gcattttcag tagagacgga gtttcaccat gttggtcagg 23220
ttggtcttga actcctgacc tcaggtgatc cgcccacctc agcctcccca agtgctggga 23280
ttacaggtgt gagccaccgt gcccagccca gccatcattt ttgaaacacg tttgagaaac 23340
agtgtcttcc tttgagggcc aaggagacat tttttttgtt tatttgtttg tttttgtgag 23400
gactagctga agggggtgat gtatattaac ctgcctactt atttgcctct tcccagagtg 23460
tgatgaatat tagggtttaa agtttctgaa gcatttgtta ataaagcccg gggctggagg 23520
tcagaagacc tggatttctc tgcatacttt tgccatcagc aagctgtgtg accttggaca 23580
gatccctttt ttgtctaaat ctttctgagt cttcttgaaa acaatgccag gttgggacag 23640
gatgattgcc aagctcccgt ccagctctaa aacactgcaa cgtatgcttc tgcaccagca 23700
ctgtccatcc tgtagatcat gcagaaattc tcttcaactt tttcctaccc ataaaatagg 23760
agcatgctta cctttttcct aatgttccag gccccgggtc tagaatattg taagtaagga 23820
agttaatgtg tatcagagcc cattatgggc cagaagttct cctcttcctt cctacacctg 23880
cttcctccct ccctccctcc ctctttccct tccttccttc catccatttg tgaagaagac 23940
atgatcaccc tcattctgag agtgaagaga cagaggctca actaatgaaa tgatttgttc 24000
aaggtcacac gggtggcaca aggcaagtgg cagaggttga atttagaccc attcctgtcc 24060
aaatgctgag tttatgtcat cgtcccgaga ccataacttt aaagatgtaa gatagtggga 24120
aaagagttga tttcaaagca cctctcagaa ggactcactt tacatcaggg gtcagcagac 24180
tcaggccaaa tccggtccat tccccgcttt tgcaaagaaa gttgtagtgg aacacagcta 24240
ggcttattga tttatggatt gccaacgtcc ttttgtgaaa cagacagctg agctgagtaa 24300
tcgtggcgca caaaacctaa aatatttact atctcgtcct ttacagaatg tttgccaatc 24360
tatggtccgg agtccaaggc tgtccatttt tcaaagaaca caaagtgaca tgagactgtc 24420
ccatgtgcag ggagccctat cattttatta tgaaaaaacg gcctttctgc tcaaatctgt 24480
tttttaaaaa gtcaacaaac agactctggg tacctgtcag gaacagtagg gagtttggtt 24540
tccattgtgc tcttcttccc aggaactcaa tgaaggggaa atagaaatct taattttggg 24600
gaaattgcac aggggaaaaa ggggagggaa tcagttacaa cactccattg cgacacttag 24660
tggggttgaa agtgacaaca gcaagggttt ctctttttgg aaatgcgagg agggtatttc 24720
cgcttctcgc agtggggcag ggtggcagac gcctagcttg ggtgagtgac tatttcttta 24780
taaaccacaa ctctgggccc gcaatggcag tccactgcct tgctgcagtc acagaatgga 24840
aatctgcaga ggcctccgca gtcacctaat cactctcctc ctcttcctgt tccattcaga 24900
gacgatctgc cgaccctctg ggagaaaatc cagcaagatg caagccttca ggtaaggcta 24960
ccccaaggag gagaaggtga gggtggatca gctggagact ggaaacatat cacagctgcc 25020
aggggctgcc aggccccaga gggcctgaga actgggtttg ggctggagag gatgtccatt 25080
attcaagaaa gaggctgtta catgcatggg cttcaggact tgtgtttcaa aatatcccag 25140
atgtggatag tgcgaccgga gggctgtctt actttcccag agactcagga acccagtgag 25200
taatagatgc atgccaagga gtgggactgc gattcaggcc tagttgaatg tgctgacaga 25260
gaagcagaga ggggcaccag gggcacagcc cgaaggccca gactgatatg ggcaaggcct 25320
gtctgtgctg acatgtcgga gggtcccact ctccagggac cttggtttcc ccgtctgtga 25380
catctgtgac atgagagtca cgataactcc ttgtgtgcct tacagggttg ttgtgaaaat 25440
taaatgcaca gataatagcg taacagtatt ccgtgcattg taaagagcct gaaaaccatt 25500
atgatttgaa aatggaatcg gctttgtgag accatcacta ttgtaaagat gtgatgctga 25560
tagaaatgac aggactgctt gtgcatgccc tctgcagtgt gacattccag cagtgaaatc 25620
atgttggggt gacttctccc ccactctgac ctttatgttt gtctgggccg aggctgcaag 25680
tcgggctctg tgggtgtatg agtgacaagt ctctcccttc cagatatggg gactgtctgc 25740
ttccctaggt tgcctctccc tgctctgatc agctagaagc tccaggagat cctcctggag 25800
gccccagcag gtgatgttta tccctccaga ctgaggctaa atctagaaac taggataatc 25860
acaaacaggc caatgctgcc atatgcaaag cactttggtt tgcctggcca cccctcgtcg 25920
agcatgtggg ctcttcagag ccacctgatg aggtgggtac agttagccac acttcacagg 25980
tgaagaggtg aggcacaggt cccaggtcag gctggccgga gctctgttta ttacgtctca 26040
cagctttgag tcctgctctc aaccagagag gccctttacc aagaagaaag gattgggacc 26100
cagaatcagg tcactggctg aggtagagag gaagccgggt tgttcccaag ggtagctgct 26160
cctgcaggac tctgagcagg tcaccagcta atggaggaaa ggctctaggg aaagaccctt 26220
ctggtctcag actcagagcg agttagctgc aaggtgttcc gtctcttgaa acttctacct 26280
aggtgctatg gtagccacta gtctcaggtg gctatttaaa tttatactta aatgaatgaa 26340
aatagaagaa aatttaaaat ccagaccctt ggtcacacta tccacattta aagaggtcaa 26400
tagccacatg tggttagtgg ccaccctatt gggcagtgca gctacagaac atttttgcat 26460
cccagaaagt tcttttggat gttgctgctc tacagcatgc tttgctgaaa cagaagtgcc 26520
ttccctggga atctcagatg ggaagcaagt aaggagggga gtcaaatgtg ggctcactgc 26580
tcaccagctg tgagggttgg gcctgcctct taaccattgt cagcctcagt cttctcatcc 26640
atgcatgccg tgggtatact aaaatactat acccctggaa gagctggatg caaatttgac 26700
aagttctggg ggacacagga aggtgccaag cacaaggctg ggcacatggt ggctgtgcac 26760
tacagctgag tccttttcct tttcagaatc tgggatgtta acc 26803
<210> 21
<211> 177
<212> PRT
<213> Homo sapiens
<400> 21
Met Glu Ile Cys Arg Gly Leu Arg Ser His Leu Ile Thr Leu Leu Leu
1 5 10 15
Phe Leu Phe His Ser Glu Thr Ile Cys Arg Pro Ser Gly Arg Lys Ser
20 25 30
Ser Lys Met Gln Ala Phe Arg Ile Trp Asp Val Asn Gln Lys Thr Phe
35 40 45
Tyr Leu Arg Asn Asn Gln Leu Val Ala Gly Tyr Leu Gln Gly Pro Asn
50 55 60
Val Asn Leu Glu Glu Lys Ile Asp Val Val Pro Ile Glu Pro His Ala
65 70 75 80
Leu Phe Leu Gly Ile His Gly Gly Lys Met Cys Leu Ser Cys Val Lys
85 90 95
Ser Gly Asp Glu Thr Arg Leu Gln Leu Glu Ala Val Asn Ile Thr Asp
100 105 110
Leu Ser Glu Asn Arg Lys Gln Asp Lys Arg Phe Ala Phe Ile Arg Ser
115 120 125
Asp Ser Gly Pro Thr Thr Ser Phe Glu Ser Ala Ala Cys Pro Gly Trp
130 135 140
Phe Leu Cys Thr Ala Met Glu Ala Asp Gln Pro Val Ser Leu Thr Asn
145 150 155 160
Met Pro Asp Glu Gly Val Met Val Thr Lys Phe Tyr Phe Gln Glu Asp
165 170 175
Glu
<210> 22
<211> 159
<212> PRT
<213> Homo sapiens
<400> 22
Met Ala Leu Glu Thr Ile Cys Arg Pro Ser Gly Arg Lys Ser Ser Lys
1 5 10 15
Met Gln Ala Phe Arg Ile Trp Asp Val Asn Gln Lys Thr Phe Tyr Leu
20 25 30
Arg Asn Asn Gln Leu Val Ala Gly Tyr Leu Gln Gly Pro Asn Val Asn
35 40 45
Leu Glu Glu Lys Ile Asp Val Val Pro Ile Glu Pro His Ala Leu Phe
50 55 60
Leu Gly Ile His Gly Gly Lys Met Cys Leu Ser Cys Val Lys Ser Gly
65 70 75 80
Asp Glu Thr Arg Leu Gln Leu Glu Ala Val Asn Ile Thr Asp Leu Ser
85 90 95
Glu Asn Arg Lys Gln Asp Lys Arg Phe Ala Phe Ile Arg Ser Asp Ser
100 105 110
Gly Pro Thr Thr Ser Phe Glu Ser Ala Ala Cys Pro Gly Trp Phe Leu
115 120 125
Cys Thr Ala Met Glu Ala Asp Gln Pro Val Ser Leu Thr Asn Met Pro
130 135 140
Asp Glu Gly Val Met Val Thr Lys Phe Tyr Phe Gln Glu Asp Glu
145 150 155
<210> 23
<211> 180
<212> PRT
<213> Homo sapiens
<400> 23
Met Ala Leu Ala Asp Leu Tyr Glu Glu Gly Gly Gly Gly Gly Gly Glu
1 5 10 15
Gly Glu Asp Asn Ala Asp Ser Lys Glu Thr Ile Cys Arg Pro Ser Gly
20 25 30
Arg Lys Ser Ser Lys Met Gln Ala Phe Arg Ile Trp Asp Val Asn Gln
35 40 45
Lys Thr Phe Tyr Leu Arg Asn Asn Gln Leu Val Ala Gly Tyr Leu Gln
50 55 60
Gly Pro Asn Val Asn Leu Glu Glu Lys Ile Asp Val Val Pro Ile Glu
65 70 75 80
Pro His Ala Leu Phe Leu Gly Ile His Gly Gly Lys Met Cys Leu Ser
85 90 95
Cys Val Lys Ser Gly Asp Glu Thr Arg Leu Gln Leu Glu Ala Val Asn
100 105 110
Ile Thr Asp Leu Ser Glu Asn Arg Lys Gln Asp Lys Arg Phe Ala Phe
115 120 125
Ile Arg Ser Asp Ser Gly Pro Thr Thr Ser Phe Glu Ser Ala Ala Cys
130 135 140
Pro Gly Trp Phe Leu Cys Thr Ala Met Glu Ala Asp Gln Pro Val Ser
145 150 155 160
Leu Thr Asn Met Pro Asp Glu Gly Val Met Val Thr Lys Phe Tyr Phe
165 170 175
Gln Glu Asp Glu
180
<210> 24
<211> 143
<212> PRT
<213> Homo sapiens
<400> 24
Met Gln Ala Phe Arg Ile Trp Asp Val Asn Gln Lys Thr Phe Tyr Leu
1 5 10 15
Arg Asn Asn Gln Leu Val Ala Gly Tyr Leu Gln Gly Pro Asn Val Asn
20 25 30
Leu Glu Glu Lys Ile Asp Val Val Pro Ile Glu Pro His Ala Leu Phe
35 40 45
Leu Gly Ile His Gly Gly Lys Met Cys Leu Ser Cys Val Lys Ser Gly
50 55 60
Asp Glu Thr Arg Leu Gln Leu Glu Ala Val Asn Ile Thr Asp Leu Ser
65 70 75 80
Glu Asn Arg Lys Gln Asp Lys Arg Phe Ala Phe Ile Arg Ser Asp Ser
85 90 95
Gly Pro Thr Thr Ser Phe Glu Ser Ala Ala Cys Pro Gly Trp Phe Leu
100 105 110
Cys Thr Ala Met Glu Ala Asp Gln Pro Val Ser Leu Thr Asn Met Pro
115 120 125
Asp Glu Gly Val Met Val Thr Lys Phe Tyr Phe Gln Glu Asp Glu
130 135 140
<210> 25
<211> 3804
<212> DNA
<213> Homo sapiens
<400> 25
taatttcagt gaaattatat aacttggtta catttaggtt acttattaca aatgaaaata 60
aatatactaa cagaagctgc ctcatatgca aatgagtaac atgttagaga ctaaaataca 120
caaaaaaatg catgtgcttt agctcatata aacaatatac aggacaactt accgggtagc 180
ataaacaacc aacaacggac tatcaaaagg aacctcgtca tctagtaact ttaaccttgt 240
tggtagatct cctttttcca tctccatata cacaggattg gaacttgcca tttctgaaat 300
ataaagcata tccttttcca aactttatct cttaggcgga aatattcaat ttatgaacat 360
gttcattcag caatcattag tcacgtacct actgtgtgga cctattatgt gcaaggccca 420
atattaagca ttacaagaga aacaaataaa agtacaagag atgtaatttc tacttttaag 480
aaccttaaaa tctaattggg aggcaaagca tattaaaagt tttaaataag acgaaggaac 540
atataattaa tacatcagtg actattactc ttaaaataat gagtatctag gcctatatat 600
agcaactgtc catataagca cacatcaaaa accaatatac atagctatca tcaatgccaa 660
gttgctcagt tttttctaga actagtattc ctactccatc ttttagaatt aaattcaatg 720
aaattaattt taaaatattt gacctttttt taaagttacc agtatagcaa atactcattc 780
tctgaagaac agaagatttg actttttgga atagccaaaa gtcacttaaa atcaagtctg 840
gtgaaaggag tgatgatcta aaactatttg tggtttaaaa aaaaaaagta tgaatctaaa 900
gtaaagtggg tttttttcca agtagttttc ttttttagtt ttttagaaac agggtctcat 960
tctgttgccc aggctggagt gcagtggtga tcactgcaac cttgaactcc tggctgtgtg 1020
tgtgtgtgtg tgtgtgtgca gcgacggggt cttcctacgt tgcctagctg cagtgcctgt 1080
gtgcacacat gcgcacatgc gtgtgtggag agacagggtc ttgctacgtt gcctagctgc 1140
aatacctggg tgtgtgcatg tgtgtgtgtg tagagacaag gtcttgaaac gttgtctagg 1200
ctggcatgtg tgtgtgtgtt tatatgtatg tgtgtgtaga cagggtcttg ctacattgca 1260
taggctggtg tgtgtgtttt tgtgtgtgta gagacggggt cttgctacgt tgcctaagct 1320
gggcaagtat ttttcaactg cctctgaaga caaatcccaa ataacagttc caaaagctgt 1380
ttcccataat tatcacatca ttagaagggt gaggcctact caccaggttc taagagccaa 1440
cattcatttt ctgatacatg cttttttaaa aaagtcattt tttccccagt ctcattgttt 1500
cccatttgac tgtgtcaggc aataagtact ttaaaggaat tcagaggagg aggccattca 1560
gaggtttggg gaagcctgat gactgcgcgg gagctaaacc agacatatcc acctaaattc 1620
aagtaagcag ccatatcact caaattgccc accatgcttc ctctagccag cactggtagt 1680
aacaactatc actctggctg atggagactc ttttctgctc ttctgtgact gggtatgatc 1740
acataataga gacagataca gtaaatttcc aatgagtaat aatgtcacac atttgaactt 1800
acctgaggag aaatagcttg tttcttattt cacacaaaag acaatctacc tcaactcaga 1860
aaaaaaaaaa ttattatgct tttaactgct atatttgaat taaagcagat ctgtaactat 1920
agatccatgt ttctagaaag ctaaaatatc tttaagtaag atgacataaa aatgtatctc 1980
tattcacttt tggtaatgaa tgaaaagttg cttaaagtct aaagtattag aaatatggca 2040
tctgttattc aagtaggatt tggaattaag aaaattcact tcttcaaaaa catgggacta 2100
tggctgcaga aagggcaatg catatagttt ttagggtatg atagctggtt tctattatat 2160
gtcaggatga catatgcgac cttccgccaa ggtagatact gcgggctatg caccaaagtc 2220
tctgaggcag acatgtaagc gagctcttca cctatattca ttcttttcct cctggacagg 2280
ttacatttcc cagtttcctt tgcagttagt tgtggctata tgacagaatt ctcatcaatg 2340
gaaatgtaca cagaagagag gtaagccact accaggccag gcctataaga cagcactttc 2400
tacatgcttt ccccagacat agcaacccaa acatgaccac atcccttaag ggaagatgga 2460
gctgaaaata atggaaggaa cttggaatgc tagaatgctg aattaccacc tgggagacag 2520
ctacccactg acctggaata cctgtcctgg actgttacat gagcaagaaa tacacttcta 2580
tttatgtatg agttacttca ttatcagata tttattacag cagtttagct acctaagatc 2640
tctctctgcc tcagactgct tatctataaa atggaataac accatctact ccaaacatta 2700
ttgtaaggat gaaatgagac aatgctgaaa agtgtttacc ataatatctg ccacacaata 2760
agtaccccat atagtatttc tgtattagta agttacatga gagattttct tcttttaata 2820
catctgcatt tataaacatt ttactttaac ctcaacttcc ccagcactgc tctaccattt 2880
tctgaatgtc attatgagag aaataaaact aatttctagg gccaggcatg gtggctcaca 2940
cctataatcc cagcattttg ggaggccaag gtgggaagac tgctttgagg tcaggatttc 3000
aagaccaacc tgggcaacac agtaagaccc catctctata aaaaaatgaa gaaatcagag 3060
ggtacagtgg cacatgcctg taatcccacg actcagaaaa ctgaggcagg aggatcgctt 3120
gaacccaagg gatcaaggct acagtgagcc atgatcacac cactgcattc cagcctgggc 3180
acagagtgag accctgtctc taaaaataaa aaatagggcc aggcacagtg gttgatgcct 3240
gtgatcccaa cactttggga ggccaaggca ggtagatcac ttgaggtcag gcgtttgaga 3300
ccagcttggc cgacatggca aaaccctgtc tctactaaaa tacaaaaatt agctgggcgt 3360
ggtggtgcac gcctgcagtc ccagctactt gggaggctga ggcaagacaa tcacttgaac 3420
ccaggaggcg gaggttgcag tgagccaaga tggcgccact gcactcaaac agaatgaaac 3480
tctgtctcaa aaaaaataaa ataaataaaa atttaaaaac taatttctta taatccagtt 3540
gtgaatttaa ccaatgtctg aaagaactat taaaagttaa aatgaatgga aaacagaata 3600
aagggttgac cagaacagat gtgattttct acttaaatct tttttttaaa ccccaaaatt 3660
caaaactgct aatgtttttt aatacgaatt tctatctttg ataaggcaat ctgagtatta 3720
cctttcaatc cttcaataaa agtatcccaa acagaagggc tattactgta actaagcttg 3780
atactctcct tcgctctttt caag 3804
<210> 26
<211> 3804
<212> DNA
<213> Homo sapiens
<220>
<221> misc_feature
<222> (2122)..(2131)
<223> n is a, c, g, or t
<400> 26
ggcttctggc tctgagtgag gtcctgctgc aaggtttcct agatgagcca ctgagactct 60
aataagatcc agtggaaata accaggctct cgtcggaata taagtcccaa gggaagctgt 120
gccagtcttg tgggcgactg cctgacttct cctttcattt cagcaccatg aagcttctca 180
cgggcctggt tttctgctcc ttggtcctgg gtgtcagcag ccgaagcttc ttttcgttcc 240
ttggcgaggc ttttgatggt aaggcttcag aaggtttgca ggatttctga agagaaacat 300
caccctggac ctgataaact ggggaaaatg atgctttcgg aaggctgctt ttgaaccaca 360
gagttgctag tgtctgcgtt gctgaggcct gccaggaact agggtttgct gggttgcctg 420
tctcgagtct ttcagagctg ctgggaatat cccctttccc cgtagtgcag cttctcagga 480
tgtgttaagt ggatggatca catttcagaa gccgctgcaa ggtgtatcaa aaacacatct 540
cctgagccgt aagggacggg gcatccagta acaacgcaca cggggtattt ttgggcttcc 600
ttaagatttg agccgctgcc ttaggttgtg ctgcccaatg tgcctgggga gctgctaaac 660
agattagaga gtcgaggatt gttgtcagtt actcagagaa agaacaatca tcctttccag 720
gagcacctga gctgtttgtt ttgcgtagaa gatgcaaaat aaggcctgca atgggtataa 780
aatgtccctc agcataaatc gcataggagt atgactaagg ctgttgactc ttctgtcttc 840
tttctccttc ctccttcgat ttcctagttg gataatgtac agggctcttt agcctcgctc 900
tgtcaggggc tcccttcctg gtttgttctg tttccattct tccttctcca gccttcttga 960
caagagctgg gaactaacgt gcctcaagcc cccacaagga ccacagcatt ttctcattta 1020
gtttcagaat gactctgtga cgcaatcttc ctctcttgga aggtgagaaa gctgatcttg 1080
gaaggtgaga aagctgagac ttagagcagc tgaagccaat gcccagggac ttactgccag 1140
tcagcaggtg gcagggcaga ggtttgagcc cggctgtgct tgaggtcagg gctcttgcca 1200
ggtagacgca tcactgacca cctcctagag gttgatggtt atgaatctca ggcacacctt 1260
ggcatcacct gaaataccca tgccttcaac tccccagcag agtctgcaga aactggcctg 1320
gggtgtggcc tgggcactgg gactttcagt ttctctctgg gtgattagaa agtgcagcca 1380
aggctcacgc ctgtaattcc agcactttgg gaggccaagg tggatgaatc acttgaggtc 1440
atgagttccg gagcagcctg gccaacatgg tgaaaccccg tctctactaa aaatactaaa 1500
atgtagccag gcgtggtggc aggcacctgt aatcccagct actcaggagg ctgaagcacg 1560
agaatcactt gaacccgaga agcagaggtt gcagtgacta gagatcgcac cagtgtcctc 1620
caacctgggt gacagagcga gactccatct aaaaaaaatg aaaaagaaag tgcagccaag 1680
gcagagcacc actgccctat tgcttcctca agcaacccac agcatcagta cagcctacta 1740
agaaagtatt tagggacttt tatgctccta acagtcactg gaactcacgt cacaatgacg 1800
tgtattccat ttgcaagaat atatacttta ggtcggggtg cggtggctca cgcctgtaat 1860
cccagcactt tgggaggcca aggcaggggg atcacgaggt caggagttcg agaccagcct 1920
gaccaacatg gtgaaatccc cgtctctact aaaaatacaa aaattagcca ggcgtgatgg 1980
cgcatgcctg taatctcagc tactcaggag gctgaggcag aagaatctct tgaacctggg 2040
aggtggaggt tgcgatgagc tgagatagca ccactgcact ccagcctggg cgacagagca 2100
agactctgtc taaaaaaaaa annnnnnnnn naaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2160
aaagaatata aactttagta gtcagggcag aagtactctg tgtctgccac ctttctcagc 2220
atcagtattc catgtcacta cctcattcat acacactcct ggatcttatc ataggcagct 2280
tcattctata gcagtggctc ttcaccaggg cacttgaaga agccaactag gataaaggaa 2340
tgtgcttctc aacccatggt atccaaggct gctatgatca caggctgaaa gcttgaagtc 2400
agtggaagat ttgtccttcc tcattcccct ctaaggtgtt gttggagtct ttatgttctc 2460
ctgatgtccc ttctgccttt cctttccttt ccaggggctc gggacatgtg gagagcctac 2520
tctgacatga gagaagccaa ttacatcggc tcagacaaat acttccatgc tcgggggaac 2580
tatgatgctg ccaaaagggg acctgggggt gcctgggctg cagaagtgat caggtaactg 2640
gagctcctgg gacgttaggg ctgggtgagc agagcttgcc tgccttggac agtcaggagg 2700
gagacgagct ccttgtggag aagttagagg ctgcggcccc tcctcctctt gccctctctc 2760
tgcctctgtg ctcagtgtga ggtctgagtg gatggtagga gtgagtgatt cctcatcctc 2820
cctctctggg tgctgttcat ccagcctagg ggtgcccagc ctggctgaat ggggtggtgc 2880
ccagtgtttt catccctcct tccttggcct ttctgggctc ctctctgagc cctcccttgg 2940
aacagggaga atgggagggt gggctattgc tcactggcct gattattaat ctccttcttg 3000
cctgccttga ttacagcgat gccagagaga atatccagag attctttggc catggtgcgg 3060
aggactcgct ggctgatcag gctgccaatg aatggggcag gagtggcaaa gaccccaatc 3120
acttccgacc tgctggcctg cctgagaaat actgagcttc ctcttcactc tgctctcagg 3180
agatctggct gtgaggccct cagggcaggg atacaaagcg gggagagggt acacaatggg 3240
tatctaataa atacttaaga ggtggaattt gtggaaactg ggtgttatac tttgtggtat 3300
agactgcctg tttagtatga aggggcgatc catgcacatc taagtgaacg tggaggctgg 3360
gtgggtggga gacgactcct gggcacacag ggcatcctgg gcatccctga ggcaaggaca 3420
tgatgagttc agtggccacc cccacaggat cccaggggct tcagcagatc ccacccctta 3480
ccccatgtga gcagctgccc agtgagtctg taggaacccg agccacattc ccagtgagtt 3540
caactgcacc ccggcacgtt ttgctagcac ctcaatggag agctccttgc ttgcagcttt 3600
ggcttgtggc acccagcaaa agcttcctgc cacccagtgg ctacagccac acactctcca 3660
gcaagattta atctcagcct tgtgaggagc cctttcccaa atttatttct ttctgtgttt 3720
tttatccctt agtagctaat ctcatgttag ccattaataa ctctctatgt taaacccttc 3780
cttttgtatc tgcggctaca ttga 3804
<210> 27
<211> 3804
<212> DNA
<213> Homo sapiens
<400> 27
gtataaatgc agaggtgcag taactgggct tttcaggata tccagatgga gttgtggtgt 60
tgttttgctt gtggttttta acgttaactt ttttttcccc tttattttaa gagaagcaca 120
aaatgaacaa actagtgagc ccagcaacat ggatggaaat tccggagatg cagactgctt 180
tcagccagca gtcaaaaggg tatgggcaaa aaaatatgaa ccatttgggg ctcaagtttc 240
tccaaatact ttatgtgact gcaagtactg tatacgctta tttcctgtga ctcagttctt 300
ctaactaaga tgttaagcat ttggcttaaa gtgtatagca ttacaaagag tatttcccca 360
gctttggctt gccagccaac tttccattga ctctagcctg ttagccattt ttattgtttt 420
ttgtttgttt gtttgtttgt tttcctcaca tgtacacata catacagatt gttcttatat 480
gtgattttgt tctctgggaa taaaatcttc attcaacaga ggcaatatga cagaaaaacc 540
gaagtttcat gtatatgatt tttcaaagaa agtgaattgg ccctcatgtt aaacctagca 600
tttcagagct gaaagtgtct tctcattaaa tattgaagaa atcatttgag ggtgtggaga 660
aggatggaca gaattagctg cttgtgtatt tattcttctc ctgcaacttt gccacgctat 720
tttgtacctc ccttcctaat tatgataaag ctttcttaga gagcagtcag gcaatgtgta 780
ttaaatgttt aaagctttac acccttagtt ctacttgtgg acatttattt cttaagaaag 840
atgtatacta agatttagat agaaatattc attacagtat cataataaag acagtagcaa 900
ggattctgtt atctgtgttg cattaataca atagagattg atgcaactgt tcattatttt 960
gaaagcatta atgataatat tgcatcaaag agttcactga acagattgta cagtacagtt 1020
ttacacacaa aaaaaatagt ttgtgagctt taaaagggcc ttcaaatgaa tatgctaaac 1080
ggttatcttt caataaagag agtatgggta agtcttaatt tctttcattt atttcagtaa 1140
tttaatgttt ttgttgttgt tattgtttgt ttttttgaga cagagtcttg ctctgtcacc 1200
caggctggag tgcagtggtg caatctcagc tcactgcaat ctcggctcac tgcaacgtct 1260
gcctcctgga ttcaagcaat tcttctgcct cagcctccgg agtagctggg attacaggcg 1320
tgcaccacca tacctagcta atttttgtat ttttagtaga gacggagttt tgccatattg 1380
gccaggctgg tgttgaactc ctggcctcat gtgatccgcc catctcagcc tcccaaagtg 1440
ctggggttac aggcatgagc cactgtgcca gcctatttca gtaacttaat gtttttacag 1500
gcatgtatta cctataaaat taataaagcc agtgaggtat ttcttttttg aactaaagca 1560
aagctaataa taagttatag agaagttaga gaagaaatct attaagtgat actttctttg 1620
tatactgttg ggctgagtac ccttgattct tggtggtgaa caagttatca gaaatttctt 1680
ggccaggagc cgtggctcac acctgtagtc ccagcacttt gggagtccaa ggtgggtgga 1740
tcacttgagg tcagaatttc tagatcagcc tggccagcat ggtgaaaccc tgtctctatt 1800
aaaaatacag aaattagcca ggcatggtga cgtgcgcctg taattccagc tacctgggag 1860
gctgaggcag caaaatcact tgaacctggc aggcggagtt cgcagtgagc tgggatcgcg 1920
tcgctgcact ccagcctgga tgacagagca agactccatc tcaaagaaaa aataaaaaag 1980
aaatttattt acttgtgtga atttttacaa tacagatgct tctcgactta aatggggcta 2040
catcccaata aactcataag ttgcaaatac tgtaaatcaa aaatgcattg aatacaccta 2100
atgtatggaa caccatagtt tagcctatcc tactttaaat gtgttccgaa cacttagatt 2160
agcctgtagt tgggcagcat tacctactat aaagtgtatt ttctaataaa atgttgaata 2220
tctcatgtaa ctcattgaat actgaaagtg aaaaacaatg tatgggtact caaaatatgg 2280
tttctctact gaatgtgaat cactttgaca ccatcataaa gttgaaaaat tccaagtcaa 2340
accattgtaa gtcaggggct atcagtattc agtggtaaat gctggctcta actattcttc 2400
caagtcagtg gttgactgct gtttattcta taaagggtta caatttatag attctctcac 2460
ttgtagaatg agagattcag aattaatagc agacagagtc cctaccttga tggagctttc 2520
atttaagtgt gaaagtcagg tgacctaaca aggccttggc ataagtttag gatttggatt 2580
gttatgggag cttgggtagg gacatgtcat aggtaaggca acagcagggg tagagataag 2640
cttgacatat gtcaaaaatc atgaagacat cagtaatcct tgaagttggc tgaaaggtat 2700
agagttgaga aagtagttaa aaaaaaaaaa gtcaggctga gtctaggtaa ggatgtgttt 2760
ctctgaggtc agatttgttc ctgtaccata aagggactat ttagaatctt aaagctggag 2820
caatttaaaa cgttaagttt tcagattgag gtcagatttg tgacttcatg tgaggtcaga 2880
tttgttcctg taccataaag ggactattta gaatcttaaa gctggagcaa tttaaaacgt 2940
taagttttca gattgacgtt ttttgaggta tagttaataa cctgaatgtt ctgattctag 3000
tcttggtagt caataagagt tgaccagatg aatttcatag ctttgtagag gatgaaatat 3060
ttcaaggctg atttgcacaa atgtttacat agatcatgta tctttcataa gtaatatgtt 3120
tgtattatta caaggctgta aaaatttaag caggttgtta atagcacagg gggtaacaga 3180
ttaataaaat taatgaataa aattactaaa agagtccaga agtaaaccca aatacgtgga 3240
ggaattaagc atatgtatga tacacatgac attttaaaaa tcagtgggaa aaggtaaatt 3300
attttacaaa tggtgttaga agcactgatt gataattttg ttaaaagaaa cttagattcc 3360
ctattttact cctaatccaa aataaattct gagtggatct aagattaagc aaaaattaag 3420
ccggaagctg agcatggtag catgtgtctg taatctccgc aatttaggag actgagtttg 3480
gctggggagg tggtgatatg cgcctagaaa aaaaaaattt tttaagccac agatgtataa 3540
gcaaaaagcg ggcaaagagg cggaattttt tttttttttt ttgatgaagt ctcacttgtc 3600
gcccaggctg gaacgcagtg gcgtgatctc agctcactgc aacctctgcc tcccgggttc 3660
aagcgattct cctgcctcag cctcccaagt agctgggatt acaggcacct gccgaggaga 3720
ggattttttt ataattaaaa caaaacaaaa caaaaaaaca ccaaactgga agataaagta 3780
tttacaacat gtaaaagact gttt 3804
<210> 28
<211> 122
<212> PRT
<213> Homo sapiens
<400> 28
Met Lys Leu Leu Thr Gly Leu Val Phe Cys Ser Leu Val Leu Gly Val
1 5 10 15
Ser Ser Arg Ser Phe Phe Ser Phe Leu Gly Glu Ala Phe Asp Gly Ala
20 25 30
Arg Asp Met Trp Arg Ala Tyr Ser Asp Met Arg Glu Ala Asn Tyr Ile
35 40 45
Gly Ser Asp Lys Tyr Phe His Ala Arg Gly Asn Tyr Asp Ala Ala Lys
50 55 60
Arg Gly Pro Gly Gly Ala Trp Ala Ala Glu Val Ile Ser Asp Ala Arg
65 70 75 80
Glu Asn Ile Gln Arg Phe Phe Gly His Gly Ala Glu Asp Ser Leu Ala
85 90 95
Asp Gln Ala Ala Asn Glu Trp Gly Arg Ser Gly Lys Asp Pro Asn His
100 105 110
Phe Arg Pro Ala Gly Leu Pro Glu Lys Tyr
115 120
<210> 29
<211> 122
<212> PRT
<213> Homo sapiens
<400> 29
Met Lys Leu Leu Thr Gly Leu Val Phe Cys Ser Leu Val Leu Gly Val
1 5 10 15
Ser Ser Arg Ser Phe Phe Ser Phe Leu Gly Glu Ala Phe Asp Gly Ala
20 25 30
Arg Asp Met Trp Arg Ala Tyr Ser Asp Met Arg Glu Ala Asn Tyr Ile
35 40 45
Gly Ser Asp Lys Tyr Phe His Ala Arg Gly Asn Tyr Asp Ala Ala Lys
50 55 60
Arg Gly Pro Gly Gly Ala Trp Ala Ala Glu Val Ile Ser Asp Ala Arg
65 70 75 80
Glu Asn Ile Gln Arg Phe Phe Gly His Gly Ala Glu Asp Ser Leu Ala
85 90 95
Asp Gln Ala Ala Asn Glu Trp Gly Arg Ser Gly Lys Asp Pro Asn His
100 105 110
Phe Arg Pro Ala Gly Leu Pro Glu Lys Tyr
115 120
<210> 30
<211> 122
<212> PRT
<213> Homo sapiens
<400> 30
Met Lys Leu Leu Thr Gly Leu Val Phe Cys Ser Leu Val Leu Gly Val
1 5 10 15
Ser Ser Arg Ser Phe Phe Ser Phe Leu Gly Glu Ala Phe Asp Gly Ala
20 25 30
Arg Asp Met Trp Arg Ala Tyr Ser Asp Met Arg Glu Ala Asn Tyr Ile
35 40 45
Gly Ser Asp Lys Tyr Phe His Ala Arg Gly Asn Tyr Asp Ala Ala Lys
50 55 60
Arg Gly Pro Gly Gly Ala Trp Ala Ala Glu Val Ile Ser Asp Ala Arg
65 70 75 80
Glu Asn Ile Gln Arg Phe Phe Gly His Gly Ala Glu Asp Ser Leu Ala
85 90 95
Asp Gln Ala Ala Asn Glu Trp Gly Arg Ser Gly Lys Asp Pro Asn His
100 105 110
Phe Arg Pro Ala Gly Leu Pro Glu Lys Tyr
115 120
<210> 31
<211> 5687
<212> DNA
<213> Homo sapiens
<400> 31
ctctctggtt gcccttaaca ttttttcctt catttcaact ttggtgggtc tgatgattat 60
gtgtcttggg gttgctcttc tcgaggagta tcttagtagt attctctgta tttcctgaat 120
ttgaatgttg gcctgtcttt ctaggttggg gaagttctcc tggataatat cctgaagagt 180
atttttcaac ttggttctat tctccttgtt actttcaggt acaccaatca aacgtagatt 240
tggtcttgtc acatagtccc atatttcttg gaggttttat tcgttccttt ttattctttt 300
ttctctagtt ttgtcttctc gctttatttc actaagttga tcttcaatct ctgatatcct 360
tgcttctgct tgattgattc agctatcccc cgctcgatat tacaaaccat gtcacgaggc 420
gtggacaccc cccatgatat ggggagtatt atcacccccc tcttccccca ctggatatta 480
caaaccatgt catagggagg tggacatccc ccacaatatg aggagtaata tcacacccct 540
ttccccgcag tggatattat gaaccatgtc acaggcggtt aaacaccccc aacgatatgg 600
ggagtaatat cacactcctc tcccccctgg atattacgaa ccatgtcatg gggggtggac 660
accctttgca atatggggag taaaatcacc cccctctccc ccaactggat attatgaacc 720
atgtcacagt gggggaaaaa tcctctgtga tatgcagagt aatatcaccc cactctcacc 780
acctggatat tacgaaccat gtcacagggg ggtggacacc ccccaagatg ggggagtaat 840
atcacctcac tctctgccac cagatattac aaactgtgtc acaggggggt gaacaacccc 900
cacaatatgg ggagcactat cacccccctc ccccagggta ttatgatcca tgtcacaggg 960
gggtggatac cacccactat atggggagta atatcacctt tctctcccgc cctggttttt 1020
atgaaccgtg tcacaggggg gtggacaccc cttgcgatat ggggagtaat atcacccccc 1080
tctccaccat ctggatatta cgaaccatgt cacagggggg tggacacccc tgcgatatgg 1140
ggaggaatat gcccctctcc ccacctggat attacaaatc atgtcacggg ggacggacat 1200
cccccacaat atggggagta atatcaccac actctcccct gctggatatt acgaaccata 1260
tcacaggcgg ctggagacac aaggcattaa caatatttcg agtaatatta tctttccctt 1320
tgaacattat gaacaatatg acagaggggt gtacacctcc tgcgatattg ggagtaatgt 1380
catcccctcc cccactggat attaggaacc atattactgg gggatgtatt cccccttcta 1440
gattgggagg aagatcatac ttgccctccc tgaatatttg aaacaatatc ataggggttt 1500
gtacactttt acgatattgg gagtaatatc atcctttctc cccctggaaa ttaggaacaa 1560
tatcacaggg gtggtgtaca cccctgcaat atttagggta atattattgt cttctcccct 1620
cgatattagg aacaatatta caaggacggt gtaaagtacc tgccaaattg ggaaaaatac 1680
tatcctctcc ctcttgtata ttagaaacaa taacacaggg ggaatgtaca cccactgcca 1740
tattgggagt aatatcatac tcgccccatc ccccagatat taggaacaat atcacagcag 1800
gggtgtacac ttttacgata ttgggagtaa tatcatactc tctccccctg gaaattagga 1860
ataatatcac agagatggtg tagaccctct gcaatactta ggataatatt atcatctccc 1920
ccctcgatat taggaacaat attacgggga gtgtaaatta cctgccaaat tggaggtaat 1980
cctctcctct ctctccctgt attttagaaa atataacaca caggaaatgt acaacactgc 2040
gatattcgga gtaatatctt cttctcccca cctggatatt aggaacaata acacggacgg 2100
ggcgtacacc cctcgcgata ttgaatgtaa tgtcatcctc tccctccctt tatattacga 2160
acaatatcac aggggggtgt acaacccctg caatattgga agtagtatca tccattctcc 2220
catgaatatt aggaacaata tcacaggggt agagtacacc ctctgcaatt tcgggagtaa 2280
catcatcctc tcgttccctg gatattataa acaacaccac gggggggtgg gggtgtacac 2340
acccttcgat attgggagta atataatcct ttccctccct atatattaga agcaatatca 2400
caggggttgg tgtaaacttc ttgcgatatg gggattaata tcacccccct ctcctgccct 2460
ggatattatg aaccatatca cagggaggtg gacacacttt gcgatatggg gagtaatatc 2520
acgcccctct cccccccgat attacgaacc atatcacaag ggagtggacc ccccccacga 2580
tatggggagt aatatcaccc ccctctcccg ccctggatat tacgaaccat atcacagggg 2640
gatggacacc ccccgcgatg cggggagtaa tgtcaccccc ttctgccccc taggatatta 2700
cgaaccatat catggacacc ctccacgata ttggaaataa tatcatcctc tcccccttgg 2760
atattaggaa caatatcaca gggggttgta cacctcctat gatattggaa gtaatatcat 2820
cctctccctc ctggatatta gcaacaatat cacagggagt gtgtacaacc ccagcgatat 2880
ttggagtaat atcaccctct caccccatgg atatgagaaa caatatcaca ggggaggtgt 2940
acatcccacg tgatattgtg tgtaatatca ttcttcccca acccctgcaa tattgtggtg 3000
taatataatt ctctcccttc ctggacatta tgaacaatat cactactagg tgatacatta 3060
ggagtaatgt atccatagga tattatgagg aatatcacag ggtgtacacc cactgtgata 3120
ttagaggtaa tatctcccta aaatattaag aagaatatct tacacccact gtgactttag 3180
aagtaatatc tccctaaaac gttacaaata acatcgcagg gtgtacactc acagtgatat 3240
taggagtaat atctccctag aatattacaa atacacatgg tgtaaaccca ctgtgacttt 3300
agaagaacta tctccctaaa ataatacaaa aatatcgcag tgtataccat aatatcccct 3360
agaatatcat aaataatatc acagggtgta cacccactgt gataatagga ataataccac 3420
cccaggatat tatgaataat gtcacaggct gtacacccac tatgacatta ggagtaatat 3480
ctccctagga cactatgaat aacatcacag attttacacc catggtgtgc acccactatg 3540
atattaggag taatatctgc acaggatata acaaataata gtacagggtg tacacatatg 3600
atatacaccc actgtgatat taggagaaat atatccctag gatattatga ataacctctc 3660
agagtacaca cacatggtat acaccctctg tggcattagg aacaataact ttctaaaaca 3720
ttacgaataa catcacagaa tgtacacaca tggtttacac ccactgtgac aggtgcaata 3780
tctcccttgg atattatgaa taacaacaaa ctatcactgt catattagga gtaatttctc 3840
cctagaatat tacaaataac atcacagggt gttcatttat ggtgcacacc cactgtgata 3900
ttaggagtaa tatctcccta ggatattact tttcatataa aagtgtgtac atccactgtg 3960
atattgggaa aaatatttct ctaggatatt atgaataata tcacagagcg tacacccact 4020
gtgatattag gagtaataat tccctgggtt attatgaata atatcacagg atgtacaacc 4080
actgtgatat taggagcaat atcttcctag gatattacaa ataatatcac agggtgtaca 4140
cccactgtga tattaaagta atttttaggt tattgtgaat aatatcacca agtgtacaaa 4200
catggtgtac actcactgtg atatcaggag taatatctca gtaaaatatt atgaataata 4260
tcacagggta tacacccact gtgatattag cagtagtatc tttgtaggat attacaaata 4320
atatcacagg gtgtatgccc actatgacat tagaagcaat atctccctag gatatcaaaa 4380
ataatatcac agggtgtaca acttctacat cccaggttct aagggattct cctgcttcag 4440
cctcctgagt ggctgggatt acagatgccc accaccacac ctggctaatt tcgtattttc 4500
agtagagatg gggtatcacc atgctggtca ggctggtctg gaacttctga cctcaggtga 4560
tccaccagcc tcggccttcc aaagtgctgg gaatacaggt gtgagccaac gtgcttggca 4620
gagagttata tattaaataa atctggaaac atagctccca tgtttgagtg tgcatttact 4680
tttatgaaga aattatgtca gaaaacctaa ggatgataat aaatatgaaa agtaactggc 4740
atgtaaaaag gtcttttgat taagaactat aaggttcgat ttcattttta gataacgtga 4800
tcctagctct tgtatagtgc ttataaatat tctacatcaa aggaatttgt tgcacagtgt 4860
cagaataaaa taaagtgtat ttcactgctt cttaattttt aaattagact gagtttgttt 4920
tcctagagag agaagaacat tttttatttt ttctgaaaag agtaggccat attttactga 4980
gatcttagat ttgttatata ttaggttttg gtcttctaac attctccagt ggattttctc 5040
taaagtaggt atgcacagaa agagttgaat agcaaaaaag taaatcatgt aataattctg 5100
agatttttgg gtttgtcaca actgagaaat attgctgagg gtgtatggtc ctcaagtgtg 5160
aaaatgttcc ttgtgaattg cttgtatccg aaatatacac acaacattaa gtcctggttt 5220
ttatctttta ttttttccaa tccttttttc ttctcaaggt gtccaagtca cacagagcca 5280
cagaatctca caggtgtctc agaattcctc ctcctgggac tctcagagga tccagaactg 5340
cagccactcc ttgctgggct gttcctatcc atgtgcctgg tcacgatgct ggggaacctg 5400
ctcatcatcc tggccgtcag ccctgactcc cacctccaca tccccatgta cttcttcctc 5460
tccaacctgt ccttgcctga cattggtttc accttggcca cggtccccaa gatgattgta 5520
gacatgcaat cacatagcag agtcatctcc catgcaggct gtctgacaca gatacctttc 5580
tttgtccttt ttgtatgtat agatgacatg ctcctgactg tgatggccta tgactgattt 5640
gtggccatct gtcaccccct gcactaccca gtcatcatga atcctca 5687
<210> 32
<211> 5687
<212> DNA
<213> Homo sapiens
<400> 32
attgaatttt atctcagagc ccacatgaag caggatcaaa gtcagtacac atgaaaacta 60
gagcccaaag actataaagc atgaaataag gatttaagct aaccctatct tgtaaggggt 120
ttgtaaagcc cagcttgcat ctgagctaca ctagcaccag gacagccact cagtaatggg 180
gtttctcaag gttattgctt ttcattcagt tgaaatgaga gtcatttctt acccttatgc 240
cctgtgagat ttcactggag gttgttcact gaaacatttt catatcattg catcaaccct 300
cttgaactca ctgtgcctgc ccccagttca gtctgtgact cacaagtacc ctgcagcaaa 360
agaaatccaa tagagggcaa atccctcacc ttaccttcct ttctaagacc tttgatgttc 420
tcatgtgtca tttcataatt gggattgtca attagtcgcc tcatctctgg tcctcacttt 480
cctctctccc agccaaactc aaccttcagc ccacacaatg gaattcaaca aaatgaggta 540
acagttttct gtgtgagtca ctctgggcaa ctctgttcac agagcactgt gaggtgagca 600
gccagaaccc aggcaagtgt ttcagccatc caagaactgg caggcagccc aagagacact 660
ctcacctgat gacagactag caggatgagt cctggaggaa atggttccca acagctgcag 720
aaggagtctc ttggctcatg cacagcaatg ctcttctcaa ttaaaaacgt tgtcattatt 780
gacactgcag tgtaaaatcc ttttacactg tgctcacatt tctacaggcc ttcacctgct 840
ctgcccatta aagacaagac ccttccatga gatgatgaca tctctaagtt actgttccac 900
ccaaacagtc ctatataatg aagagaaaaa ttttgctggc cctcaaaagg caaacacaag 960
gagaaagatt tccacaagct gtttctcttt gctgagcact tagaggaaaa ctgtaagtgg 1020
ttggaagaag gctttgtttt ctacaagact tttagttatt cctcagaaat tttcctgctt 1080
attcccagag gaggtcatct cttagatgct gtcagtcaga tagggattgg cagccaagca 1140
gaggtgctca gagaggtttc caactaatgt ggccagcgga aaactgccaa agaagcaggg 1200
atccttagga caaataaact ggaagatatt ttggggataa aaataaatcc ttttgaaaat 1260
gaaagatgga gagatgctgt atatacaaat tgccctgttc tgaacaatgt tgtcactagg 1320
actggccctg gagaccaatg atacaaacca aaatgttctc agacatgctt tgatggtctt 1380
tttctccaaa gttatctatt ctgtttccat ttcattctca caggacttgc catggggttc 1440
tcataagatt tcacattggt cataatccag gtggccctgg actgcaacct ctgagttggc 1500
aacatcagaa taggaattac gaaaaaccaa tttaaagtta aatacagaca caggcaaaag 1560
agagatgggt tgtcgaagct agtgcctagg tggacactgc ctcacatttt taattccaga 1620
agccatcagt actgagtgtc agatctcatt agtcaaacac agtgatcagg aatcctgttt 1680
tcctggagga tttccttgag ggagggacca ctcaagagtc tgaaatattt cacgtcatag 1740
agtatggatc tcaccccaac acccaatcag aaaataggga gaactggaaa ccaaaattcc 1800
ccctcccgct gtggaaggat gaaaaccaga gtgttggagt tctgtcctga taatggagca 1860
gacagctagg cagcaatcaa tgagggccag tacaggaatt cagtgctaag tattgggtca 1920
taacagaagt agggaaggta ttaatccagt gctatatgag gatcctggga cactggctcc 1980
tagtaatcta gttataacta ttagcaaaaa agaaaaaaaa aatcagtgat gtgaagagat 2040
ggcctaaagg agctccagca atatagctaa gcagctggca agtggtctga ggagcattgc 2100
aattccaggc ctcctaaggt ggcagtacgg gcactggtaa gacattctgc tgtggtgaaa 2160
ctagtttacc atagaggatt cacaattaaa ataggcaaac aggaaatgca agacagaggc 2220
taacaaaggg tttttttttt ggtggggggg agttgtttgt ttgtttgttt gttttctgag 2280
acggagtctt cctctgttgc ccaggctgga atgcagtggc acgatctcag ctcactacaa 2340
cctctgcctc ccaggttcaa gcagttcttc tgcctcagcc tcccaggttc aagcagttct 2400
tctgcctcag cctcccaagt acctgggact atagctgtgt gccaccacat tgactaattt 2460
ttgtactttt agtacagact gggtttcacc atgttggcca ggctggtgtc gaactcctga 2520
cctcaagtaa tccccccgcc ttggcctccc aaagtgctgg aattacaggc atgagccacc 2580
acacctggcc agtttttggt aattcttaaa gaactcaatg agcaacactc aaacaaccat 2640
aaagactata gagctcatgg ttgaatttta gatagctaaa cagacaggag tttttgtaag 2700
ttttgtaagt cttgctcatc cttccctctt ccatcctcta tctcaactat tctgtctacc 2760
attaaagcac cttagacctt gagtttggca atgcaacaag tgtgtgctca acacgaaata 2820
ggtaattcaa tagcaaagcc ctaaaacagc ctggcttgat tatttctcag ggcatgcagt 2880
tcctttgaag caggatcatt ttaataataa taataataga aataataata gaaattgaag 2940
acaattattt cacaatttcc atacacctaa gagctataca tatgaatgat aatgcataat 3000
tgtaaagcat gcatattaca ggtaataaat atgttagcta attataaaca atgcccattt 3060
tcatatagtt tatccttgcc aaataaaact gtaaaaaaaa gacacctttc aaatgctgct 3120
aaggagtaat acctgaatga ggttgattta atggagtctt agttcctgca tgtgttctaa 3180
ttgaatagac tatgtagtaa ttcccttaca tacccatcca tgtccaagaa cagtgaagat 3240
ctttatttaa tatgaattat tgcagatgat tagcacagtc tagccaaacc attccagtaa 3300
ttgtttttac ttgttatatt aatatataaa ttctcaaagg atataacagt gatgttgggt 3360
gaatttcact gaatgatagc tcaaacacct gaaatattga ctaagaaaac taatttatca 3420
atactgataa tcaattttaa tatgttaatt gattgtaata caggattctg tggttcaaaa 3480
aaaaagaaca agcaaaaaaa ctttcttcca tttccaaata ccaattaata gatctctact 3540
tccccttgga tttcttctta ccacctacca cctccaatct tcattctttc ctcacaaact 3600
aaacataaaa gttacctaca aagcatagaa tctgtgttaa aggatattct tgcttgtttt 3660
aagtccaaaa ttaaacagct ctgaattatt aaaaagcaca tgaattcaaa tgtcctattc 3720
taataagaaa atggtttaca tttctctatg ttcaaggaaa aaaatagtca agggtgtaca 3780
agtggggtaa aaattatttc cagtaggtta tgtgatttaa gttatagaaa cgaaccaggc 3840
aattcaatta aatgtcatgg aaagtaggtt ttttcttttc ctcttttttt ctaatatgta 3900
cactttgtga gaagataaat ccatagtgtg ataatttgtc cactgggtcc atcagacact 3960
ggagacagct tcctaagaat tataaggctt ctaaaggctt ctaaagccta aattgcctag 4020
agcattttgt gtgccaggca ctttgctagg tgccctaggg atgcaagaag tataaatgtt 4080
ttatgagaat acaggctgga aatgtattct tgattattcc tgtggaattt ctaggcagaa 4140
aagagtctaa tggggtatag gtatattttc tcaacacaat tttctgagcc tttaccagat 4200
gcagttctat ggtttgaatg tgtccctcag agtttgtgtg ttggaaactt aatccccaat 4260
gcaataatgt tggtgaggcc taatgaaagc ctaataatgt aggtgaggcc taatgagagc 4320
tgtttagacc atgaaggctc ttccctcatg gatggattaa tgctgttatg gtgggaatgg 4380
gttcattatt actggggtgg gttcataata aaaagatgag tttggcctgc tattctctct 4440
ctttctcatc ctctcttcca ccatgggatg acacagcaag aaggcccctg caagatgccc 4500
tcccctcagt attggacttc acagcctcca ggaccataag ccaataaatt tttgttcatt 4560
ataaatttcc cagtctgtgg tattctgtta tagcaacact caatttatgc attacttcca 4620
gattcttatg gctataccta cttctcacag tttgtattca cccctccttc aaccaagtac 4680
ccttaacaca gttcccatag tcacaaagcc aggtcactga agctgccctc tctccaacca 4740
cacacatata gatcaaatga ccccagacat agagctgatt gagaaggagg gaccagtacg 4800
agctctgctt ccccagcagc ttcctggaaa gaagaggcaa tacaacccaa cccaaaagtg 4860
caagagaagt aacacctcat gggatgagct taattaatca atgggagagg acactagaag 4920
acactagagg atctcccttc ctccctttct ttcccacttc accccctcca gtctctgaac 4980
catgagctat ttcaaaggtg cagtaatgct atatttggct tctctgaaga tatcctatga 5040
ggccaagtca tcagctttgt tcattatcta agagtggtgg ccagctcacc agcacttccc 5100
atcatgtttg ccctccctct ttcccttgtg ttacttccca ttttccctta cttctgcttt 5160
cttggcatta aattctactc tgcaatgtta ggatataagt ttttgcctca gattctgttt 5220
tctaggaaac ccatgctaag acaacactgg cagtggccct ggaaaagtaa acctcatgat 5280
ggatttggag ttggattgtt cactgatctg aaggacagag gactccactt aagtggtaag 5340
cagtgtagct atgaactctg ccacgcaggc ctcacaatta ctgaggcttc ttttacctgt 5400
ggttaactgg gacacagaac agcaggaaat tgagtgtaga ggttatcaag tagctgcttc 5460
acttaattgg tataatttta tggagttaac ctggtttaga gtccagagaa cattccacat 5520
agcctagaaa gggtagttat ttgtccttac cataatcaag tcatactttg aatatgagtt 5580
ttccttccct gttcagcacc acttctctta gacttaagaa tgcctgatct gttgatatta 5640
tgtcccatgt aacattgcct gagacaaaga tatccatgta ccttaaa 5687
<210> 33
<211> 140
<212> PRT
<213> Homo sapiens
<400> 33
Met Gly Phe Gln Lys Phe Ser Pro Phe Leu Ala Leu Ser Ile Leu Val
1 5 10 15
Leu Leu Gln Ala Gly Ser Leu His Ala Ala Pro Phe Arg Ser Ala Leu
20 25 30
Glu Ser Ser Pro Ala Asp Pro Ala Thr Leu Ser Glu Asp Glu Ala Arg
35 40 45
Leu Leu Leu Ala Ala Leu Val Gln Asp Tyr Val Gln Met Lys Ala Ser
50 55 60
Glu Leu Glu Gln Glu Gln Glu Arg Glu Gly Ser Ser Leu Asp Ser Pro
65 70 75 80
Arg Ser Lys Arg Cys Gly Asn Leu Ser Thr Cys Met Leu Gly Thr Tyr
85 90 95
Thr Gln Asp Phe Asn Lys Phe His Thr Phe Pro Gln Thr Ala Ile Gly
100 105 110
Val Gly Ala Pro Gly Lys Lys Arg Asp Met Ser Ser Asp Leu Glu Arg
115 120 125
Asp His Arg Pro His Val Ser Met Pro Gln Asn Ala
130 135 140
<210> 34
<211> 128
<212> PRT
<213> Homo sapiens
<400> 34
Met Gly Phe Gln Lys Phe Ser Pro Phe Leu Ala Leu Ser Ile Leu Val
1 5 10 15
Leu Leu Gln Ala Gly Ser Leu His Ala Ala Pro Phe Arg Ser Ala Leu
20 25 30
Glu Ser Ser Pro Ala Asp Pro Ala Thr Leu Ser Glu Asp Glu Ala Arg
35 40 45
Leu Leu Leu Ala Ala Leu Val Gln Asp Tyr Val Gln Met Lys Ala Ser
50 55 60
Glu Leu Glu Gln Glu Gln Glu Arg Glu Gly Ser Arg Ile Ile Ala Gln
65 70 75 80
Lys Arg Ala Cys Asp Thr Ala Thr Cys Val Thr His Arg Leu Ala Gly
85 90 95
Leu Leu Ser Arg Ser Gly Gly Val Val Lys Asn Asn Phe Val Pro Thr
100 105 110
Asn Val Gly Ser Lys Ala Phe Gly Arg Arg Arg Arg Asp Leu Gln Ala
115 120 125
<210> 35
<211> 141
<212> PRT
<213> Homo sapiens
<400> 35
Met Gly Phe Gln Lys Phe Ser Pro Phe Leu Ala Leu Ser Ile Leu Val
1 5 10 15
Leu Leu Gln Ala Gly Ser Leu His Ala Ala Pro Phe Arg Ser Ala Leu
20 25 30
Glu Ser Ser Pro Ala Asp Pro Ala Thr Leu Ser Glu Asp Glu Ala Arg
35 40 45
Leu Leu Leu Ala Ala Leu Val Gln Asp Tyr Val Gln Met Lys Ala Ser
50 55 60
Glu Leu Glu Gln Glu Gln Glu Arg Glu Gly Ser Ser Leu Asp Ser Pro
65 70 75 80
Arg Ser Lys Arg Cys Gly Asn Leu Ser Thr Cys Met Leu Gly Thr Tyr
85 90 95
Thr Gln Asp Phe Asn Lys Phe His Thr Phe Pro Gln Thr Ala Ile Gly
100 105 110
Val Gly Ala Pro Gly Lys Lys Arg Asp Met Ser Ser Asp Leu Glu Arg
115 120 125
Asp His Arg Pro His Val Ser Met Pro Gln Asn Ala Asn
130 135 140
<210> 36
<211> 116
<212> PRT
<213> Homo sapiens
<400> 36
Ala Pro Phe Arg Ser Ala Leu Glu Ser Ser Pro Ala Asp Pro Ala Thr
1 5 10 15
Leu Ser Glu Asp Glu Ala Arg Leu Leu Leu Ala Ala Leu Val Gln Asp
20 25 30
Tyr Val Gln Met Lys Ala Ser Glu Leu Glu Gln Glu Gln Glu Arg Glu
35 40 45
Gly Ser Ser Leu Asp Ser Pro Arg Ser Lys Arg Cys Gly Asn Leu Ser
50 55 60
Thr Cys Met Leu Gly Thr Tyr Thr Gln Asp Phe Asn Lys Phe His Thr
65 70 75 80
Phe Pro Gln Thr Ala Ile Gly Val Gly Ala Pro Gly Lys Lys Arg Asp
85 90 95
Met Ser Ser Asp Leu Glu Arg Asp His Arg Pro His Val Ser Met Pro
100 105 110
Gln Asn Ala Asn
115
<210> 37
<211> 187
<212> PRT
<213> Homo sapiens
<400> 37
Thr Ser Glu Glu Thr Ile Ser Thr Val Gln Glu Lys Gln Gln Asn Ile
1 5 10 15
Ser Pro Leu Val Arg Glu Arg Gly Pro Gln Arg Val Ala Ala His Ile
20 25 30
Thr Gly Thr Arg Gly Arg Ser Asn Thr Leu Ser Ser Pro Asn Ser Lys
35 40 45
Asn Glu Lys Ala Leu Gly Arg Lys Ile Asn Ser Trp Glu Ser Ser Arg
50 55 60
Ser Gly His Ser Phe Leu Ser Asn Leu His Leu Arg Asn Gly Glu Leu
65 70 75 80
Val Ile His Glu Lys Gly Phe Tyr Tyr Ile Tyr Ser Gln Thr Tyr Phe
85 90 95
Arg Phe Gln Glu Glu Ile Lys Glu Asn Thr Lys Asn Asp Lys Gln Met
100 105 110
Val Gln Tyr Ile Tyr Lys Tyr Thr Ser Tyr Pro Asp Pro Ile Leu Leu
115 120 125
Met Lys Ser Ala Arg Asn Ser Cys Trp Ser Lys Asp Ala Glu Tyr Gly
130 135 140
Leu Tyr Ser Ile Tyr Gln Gly Gly Ile Phe Glu Leu Lys Glu Asn Asp
145 150 155 160
Arg Ile Phe Val Ser Val Thr Asn Glu His Leu Ile Asp Met Asp His
165 170 175
Glu Ala Ser Phe Phe Gly Ala Phe Leu Val Gly
180 185
<210> 38
<211> 168
<212> PRT
<213> Homo sapiens
<400> 38
Val Arg Glu Arg Gly Pro Gln Arg Val Ala Ala His Ile Thr Gly Thr
1 5 10 15
Arg Gly Arg Ser Asn Thr Leu Ser Ser Pro Asn Ser Lys Asn Glu Lys
20 25 30
Ala Leu Gly Arg Lys Ile Asn Ser Trp Glu Ser Ser Arg Ser Gly His
35 40 45
Ser Phe Leu Ser Asn Leu His Leu Arg Asn Gly Glu Leu Val Ile His
50 55 60
Glu Lys Gly Phe Tyr Tyr Ile Tyr Ser Gln Thr Tyr Phe Arg Phe Gln
65 70 75 80
Glu Glu Ile Lys Glu Asn Thr Lys Asn Asp Lys Gln Met Val Gln Tyr
85 90 95
Ile Tyr Lys Tyr Thr Ser Tyr Pro Asp Pro Ile Leu Leu Met Lys Ser
100 105 110
Ala Arg Asn Ser Cys Trp Ser Lys Asp Ala Glu Tyr Gly Leu Tyr Ser
115 120 125
Ile Tyr Gln Gly Gly Ile Phe Glu Leu Lys Glu Asn Asp Arg Ile Phe
130 135 140
Val Ser Val Thr Asn Glu His Leu Ile Asp Met Asp His Glu Ala Ser
145 150 155 160
Phe Phe Gly Ala Phe Leu Val Gly
165
Claims (74)
- 컴퓨터 판독 가능 매체상에 저장된 컴퓨터 프로그램 명령들을 실행하기 위한 하드웨어 프로세서를 작동시키는 단계를 포함하는, 대상체(subject)의 혈액 내 복수의 폴리펩타이드의 발현값을 포함하는 생물학적 데이터를 분석하는 방법으로,
상기 컴퓨터 프로그램 명령들은 한 방향의 좌표 δ1로 정의되는 곡선상의 한 점에서 계산되는, 곡선의 한 세그먼트 및 한 축 간의 거리를 계산하는 컴퓨터 프로그램 명령들; 및 상기 거리를 대상체의 박테리아성 감염 유무 또는 박테리아성 감염 가능성과 연관시키는 컴퓨터 프로그램 명령들을 포함하고,
상기 좌표는 상기 발현값들의 조합에 의해 정의되고, 상기 세그먼트의 적어도 90%는 하한선 f(δ1)-ε0과 상한선 f(δ1)+ε1 사이에 존재하고, 상기 f(δ1)는 1/(1+exp(δ1))과 동일하고 ε0 및 ε1은 각각 0.5 미만인 것인, 생물학적 데이터를 분석하는 방법. - 제 1항에 있어서,
상기 발현값은 자동화된 ELISA, 자동화된 면역분석법(immunoassay), 및 자동화된 기능적 분석법(functional assay)으로 이루어진 군으로부터 선택되는 하나 이상의 자동화된 분석법으로 수행되는 측정 시스템에 의해 측정되고, 상기 방법은 상기 측정 시스템으로부터 상기 생물학적 데이터를 수신하는 단계를 포함하는, 생물학적 데이터를 분석하는 방법. - 제 2항에 있어서,
상기 생물학적 데이터를 수신하는 단계는 네트워크 인터페이스에 의한 인터넷 네트워크를 통해서 이루어지는, 생물학적 데이터를 분석하는 방법. - 제 1항 내지 제 3항 중 어느 한 항에 있어서,
상기 발현값의 조합은 상기 발현값의 선형 결합(linear combination)을 포함하는, 생물학적 데이터를 분석하는 방법. - 제 1항 내지 제4항 중 어느 한 항에 있어서,
상기 발현값의 조합은 상기 발현값들 중 하나 이상과 관련된 하나 이상의 비 선형항을 포함하는, 생물학적 데이터를 분석하는 방법. - 제 1항 내지 제 5항 중 어느 한 항에 있어서,
상기 거리에 기초하여 상기 가능성을 얻는 단계, 상기 가능성을 미리 결정된 임계값과 비교하는 단계 및 상기 가능성이 미리 결정된 임계값 이상일 때 상기 대상체의 박테리아성 감염을 치료하는 단계를 더 포함하는, 생물학적 데이터를 분석하는 방법. - 컴퓨터 판독 가능 매체 상에 저장된 컴퓨터 프로그램 명령들을 실행하기 위한 하드웨어 프로세서를 작동시키는 단계를 포함하는, 대상체(subject)의 혈액 내 복수의 폴리펩타이드의 발현값을 포함하는 생물학적 데이터를 분석하는 방법으로,
상기 컴퓨터 프로그램 명령들은 제1 방향의 제 1좌표 δ0 및 제 2 방향의 제 2 좌표 δ1으로 정의되는 상기 평면상의 한 점에서 계산되는, 곡면의 한 세그먼트 및 제 1 방향 및 제 2 방향에 의해 정의되는 평면 간의 제 1 거리를 계산하는 컴퓨터 프로그램 명령들; 및 상기 제 1 거리를 대상체의 박테리아성 감염 여부 또는 박테리아성 감염 가능성과 연관시키는 컴퓨터 프로그램 명령들을 포함하고,
상기 좌표는 상기 발현값들의 다른 조합에 의해 정의되고, 상기 세그먼트의 적어도 90%는 하한면 f(δ0,δ1)-ε0에서 상한면 f(δ0,δ1)+ε1 사이에 존재하고, 상기 f(δ0,δ1) 는 exp(δ1)/(1+exp(δ0)+exp(δ1))과 동일하고 ε0 및 ε1은 각각 0.5 미만인 것인, 생물학적 데이터를 분석하는 방법. - 제 7항에 있어서,
상기 발현값은 자동화된 ELISA, 자동화된 면역분석법(immunoassay), 및 자동화된 기능적 분석법(functional assay)으로 이루어진 군으로부터 선택되는 하나 이상의 자동화된 분석법으로 수행되는 측정 시스템에 의해 측정되고, 상기 방법은 상기 측정 시스템으로부터 상기 생물학적 데이터를 수신하는 단계를 포함하는, 생물학적 데이터를 분석하는 방법. - 제 8항에 있어서,
상기 수신하는 단계는 네트워크 인터페이스에 의한 인터넷 네트워크를 통해서 이루어지는, 생물학적 데이터를 분석하는 방법. - 제 7항 내지 제 9항 중 어느 한 항에 있어서,
상기 좌표들 중 하나 이상에 대해, 상기 발현값의 조합은 상기 발현값의 선형 결합 (linear combination)을 포함하는, 생물학적 데이터를 분석하는 방법. - 제 7항 내지 제 10항 중 어느 한 항에 있어서,
상기 좌표들 중 하나 이상에 대해, 상기 발현값의 조합은 상기 발현값들 중 하나 이상과 관련된 하나 이상의 비선형항을 포함하는, 생물학적 데이터를 분석하는 방법. - 제 7항 내지 제 11항 중 어느 한 항에 있어서,
상기 거리에 기초하여 상기 가능성을 얻는 단계, 상기 가능성을 미리 결정된 임계값과 비교하는 단계 및 상기 가능성이 미리 결정된 임계값 이상일 때 상기 대상체의 박테리아성 감염을 치료하는 단계를 더 포함하는, 생물학적 데이터를 분석하는 방법. - 제 7항 내지 제12항 중 어느 한 항에 있어서,
제 2 곡면의 한 세그먼트 및 상기 평면 간의 제2 거리를 계산하는 단계; 및
상기 제 2 거리를 대상체의 바이러스성 감염 여부 또는 바이러스성 감염 가능성과 연관시키는 단계;를 더 포함하는, 생물학적 데이터를 분석하는 방법으로,
상기 제 2 곡면의 세그먼트의 적어도 90%는 하한면 g(δ0,δ1)-ε2에서 상한면 g(δ0,δ1)+ε3 사이에 존재하고, 상기 g(δ0,δ1) 는 exp(δ0)/(1+exp(δ0)+exp(δ1))과 동일하고 ε2 및 ε3은 각각 0.5 미만인 것인, 생물학적 데이터를 분석하는 방법. - 제 13항에 있어서, 상기 제 2 거리에 기초하여 상기 가능성을 얻는 단계, 상기 가능성을 미리 결정된 제 2 임계값과 비교하는 단계 및 상기 가능성이 미리 결정된 제 2 임계값 이상일 때 상기 대상체의 바이러스성 감염을 치료하는 단계를 더 포함하는, 생물학적 데이터를 분석하는 방법.
- 제 13항에 있어서,
제 1 거리에 기초하여 대상체의 박테리아성 감염 가능성을 얻는 단계, 제 2 거리에 기초하여 대상체의 바이러스성 감염 가능성을 얻는 단계, 상기 각 가능성을 각각의 미리 결정된 임계값과 비교하는 단계, 및 상기 각 가능성이 각각의 미리 결정된 임계값 이하인 경우 대상체를 비 감염성 질병을 가질 것으로 결정하는 단계를 더 포함하는, 생물학적 데이터를 분석하는 방법. - 컴퓨터 판독 가능 매체 상에 저장된 컴퓨터 프로그램 명령들을 실행하기 위한 하드웨어 프로세서를 작동시키는 단계를 포함하는, 대상체(subject)의 혈액 내 복수의 폴리펩타이드의 발현값을 포함하는 생물학적 데이터를 분석하는 방법으로,
상기 컴퓨터 프로그램 명령들은 제1 방향의 제 1좌표 δ0 및 제 2 방향의 제 2 좌표 δ1으로 정의되는 상기 평면상의 한 점에서 계산되는, 곡면의 한 세그먼트 및 제 1 방향 및 제 2 방향에 의해 정의되는 평면 간의 거리를 계산하는 컴퓨터 프로그램 명령들; 및 상기 거리를 대상체의 바이러스성 감염 여부 또는 바이러스성 감염 가능성과 연관시키는 컴퓨터 프로그램 명령들을 포함하고,
상기 좌표는 상기 발현값들의 다른 조합에 의해 정의되고, 상기 세그먼트의 적어도 90%는 하한면 g(δ0,δ1)-ε0에서 상한면 g(δ0,δ1)+ε1 사이에 존재하고, 상기 g(δ0,δ1) 는 exp(δ0)/(1+exp(δ0)+exp(δ1))과 동일하고 ε0 및 ε1은 각각 0.5 미만인 것인, 생물학적 데이터를 분석하는 방법. - 제 16항에 있어서,
상기 발현값은 자동화된 ELISA, 자동화된 면역분석법(immunoassay), 및 자동화된 기능적 분석법(functional assay)으로 이루어진 군으로부터 선택되는 하나 이상의 자동화된 분석법으로 수행되는 측정 시스템에 의해 측정되고, 상기 방법은 상기 측정 시스템으로부터 상기 생물학적 데이터를 수신하는 단계를 포함하는, 생물학적 데이터를 분석하는 방법. - 제 17항에 있어서,
상기 생물학적 데이터를 수신하는 단계는 네트워크 인터페이스에 의한 인터넷 네트워크를 통해서 이루어지는, 생물학적 데이터를 분석하는 방법. - 제 1항 내지 제 18항 중 어느 한 항에 있어서,
상기 복수의 폴리펩타이드 각각은 CRP, IP-10, TRAIL, IL1ra, PCT 및 SAA로 이루어진 군으로부터 선택되는 것인, 생물학적 데이터를 분석하는 방법. - 제 1항 내지 제 18항 중 어느 한 항에 있어서,
상기 복수의 폴리펩타이드는 3개 이상의 폴리펩타이드를 포함하는 것인, 생물학적 데이터를 분석하는 방법. - 제 1항 내지 제 18항 중 어느 한 항에 있어서,
상기 복수의 폴리펩타이드는 CRP, IP-10, TRAIL, IL1ra, PCT 및 SAA로 이루어진 군으로부터 선택된 3개 이상의 폴리펩타이드를 포함하는 것인, 생물학적 데이터를 분석하는 방법. - 제 1항 내지 제 18항 중 어느 한 항에 있어서,
상기 복수의 폴리펩타이드는 적어도 CRP 및 TRAIL를 포함하는 것인, 생물학적 데이터를 분석하는 방법. - 제 1항 내지 제 18항 중 어느 한 항에 있어서,
상기 복수의 폴리펩타이드는 적어도 CRP, TRAIL 및 IP-10를 포함하는 것인, 생물학적 데이터를 분석하는 방법. - 제 1항 내지 제 23항 중 어느 한 항에 있어서,
상기 가능성의 결과를 생성하는 단계를 더 포함하고, 상기 결과는 문자로 표현되는 것인, 생물학적 데이터를 분석하는 방법. - 제 1항 내지 제 23항 중 어느 한 항에 있어서,
상기 가능성의 결과를 생성하는 단계를 더 포함하고, 상기 결과는 그래프로 표현되는 것인, 생물학적 데이터를 분석하는 방법. - 제 1항 내지 제23항 중 어느 한 항에 있어서,
상기 가능성의 결과를 생성하는 단계를 더 포함하고, 상기 결과는 컬러 인덱스(color index)로 표현되는 것인, 생물학적 데이터를 분석하는 방법. - 제 1항 내지 제 15항 중 어느 한 항에 있어서,
상기 혈액 샘플은 전혈인 것인, 생물학적 데이터를 분석하는 방법. - 제 1항 내지 제 15항 중 어느 한 항에 있어서,
상기 혈액 샘플은 전혈의 분획인 것인, 생물학적 데이터를 분석하는 방법. - 제 28항에 있어서,
상기 혈액의 분획은 혈청 또는 혈장을 포함하는 것인, 생물학적 데이터를 분석하는 방법. - 제 1항 내지 제 29항 중 어느 한 항에 있어서,
상기 발현값을 결정하는 단계를 더 포함하고, 상기 하나 이상의 발현값은 전기영동적으로 또는 면역화학적으로 결정되는 것인, 생물학적 데이터를 분석하는 방법. - 제 30항에 있어서,
상기 면역화학적 결정은 유세포 분석법(flow cytometry), 방사 면역 분석법(radioimmunoassay), 면역 형광법(immunofluorescence) 또는 효소 결합 면역 흡착 분석법(enzyme-linked immunosorbent assay)에 의해 수행되는 것인, 생물학적 데이터를 분석하는 방법. - 제 1항 내지 제 31항 중 어느 한 항에 있어서,
상기 계산하는 단계 및 연관시키는 단계는 대상체로부터 떨어진 컴퓨터에 의해 실행되는 것인, 생물학적 데이터를 분석하는 방법. - 제 1항 내지 제 31항 중 어느 한 항에 있어서,
상기 상기 계산하는 단계 및 연관시키는 단계는 대상체에 근접한 컴퓨터에 의해 실행되는 것인, 생물학적 데이터를 분석하는 방법. - 제 1항 내지 제 31항 중 어느 한 항에 있어서,
상기 계산하는 단계 및 연관시키는 단계는 클라우드 컴퓨터 설비의 클라우드 컴퓨터 리소스에 의해 실행되는 것인, 생물학적 데이터를 분석하는 방법. - 하드웨어 프로세서에 의해 판독될 때, 하드웨어 프로세서로 하여금 미지의 질병을 가진 대상체의 혈액 내 복수의 폴리펩타이드들의 발현값을 수신하도록 하는 프로그램 명령이 저장된 컴퓨터 판독 가능 매체를 포함하고, 제 1항 내제 제 10항, 제13항 및 제15항 내지 제34항 중 어느 한 항에 따른 방법으로 실행되는 것인, 컴퓨터 소프트웨어 제품.
- 자동화된 ELISA, 자동화된 면역 분석법 및 자동화된 기능 분석법으로 이루어진 그룹으로부터 선택되는 하나 이상을 수행하는 측정 시스템에 의해 미지의 질병을 가진 대상체의 혈액 샘플에서 측정된 복수의 폴리펩타이드의 발현값을 수신하는 유저 및/또는 네트워크 인터페이스; 및
제 35항의 컴퓨터 소프트웨어 제품을 저장하는 컴퓨터 판독 가능 매체를 가진 하드웨어 프로세서를 포함하는, 생물학적 데이터를 분석하는 시스템. - 미지의 질병을 가진 대상체의 혈액 내 복수의 폴리펩타이드의 발현값을 측정하기 위해 고안된 제 1 구획;
제 35항의 컴퓨터 소프트웨어 제품을 저장하는 컴퓨터 판독 가능 매체를 가진 하드웨어 프로세서를 포함하는 제 2 구획; 및
디스플레이 장치;
를 포함하는 생물학적 데이터를 분석하는 시스템으로,
상기 하드웨어 프로세서는 상기 제 1구획으로부터 계산된 발현값을 받아 상기 디스플레이 장치에 분석 결과를 내보내도록 고안된 것인, 생물학적 데이터를 분석하는 시스템. - 제 37항에 있어서,
상기 제 1구획, 제 2 구획 및 디스플레이 장치는 휴대용 장치(hand-held device)의 본체에 장착되거나 일체형으로 구성되는 것인, 생물학적 데이터를 분석하는 시스템. - 컴퓨터 판독 가능 매체에 저장된 컴퓨터 프로그램 명령을 수행하는 하드웨어 프로세서를 작동시키는 단계를 포함하는, 데이터세트를 분석하는 방법으로,
상기 컴퓨터 프로그램 명령은 하기 명령들을 포함하고:
다수의 대상체의 혈액 샘플 내에서 미지의 질병을 가진 대상체의 혈액 내 복수의 폴리펩타이드의 발현값에 기초하여 분류된 그룹을 포함하는 데이터에 엑세스하기 위한 컴퓨터 프로그램 명령으로, 상기 분류된 그룹은 박테리아성 감염, 바이러스성 감염 및 비-바이러스성, 비-박테리아성 질병을 포함하고; 및
특정 대상체의 박테리아성 감염 가능성을 나타내는 하나 이상의 제 1 확률 분류 함수 f(δ0,δ1)를 제공하기 위해 상기 분류 그룹들을 분석하는 컴퓨터 프로그램 명령으로, 상기 제 1 분류 함수는 제 1 좌표 δ0 및 제 2 좌표 δ1의 함수이고, 상기 좌표는 상기 발현값들의 다른 조합으로 정의된다. - 제 39항에 있어서,
특정 대상체의 바이러스성 감염 가능성을 나타내는 제 2 분류 함수 g(δ0,δ1)를 계산하는 단계를 더 포함하고, 상기 제 2 분류 함수는 상기 제 1 및 제 2 좌표의 함수인 것인, 데이터세트를 분석하는 방법. - 제 39항 또는 제 40항에 있어서,
특정 대상체가 비 바이러스성, 비 박테리아성 질병을 가질 가능성을 나타내는 제 3 분류 함수 h(δ0,δ1)를 계산하는 단계를 더 포함하고, 상기 제 3 분류 함수는 제 1 및 제 2 좌표의 함수인 것인, 데이터세트를 분석하는 방법. - 제 39항 내지 제 41항 중 어느 한 항에 있어서,
상기 좌표들 중 하나 이상에 대해, 상기 발현값의 조합은 상기 발현값들의 선형 결합을 포함하는 것인, 데이터세트를 분석하는 방법. - 제 39항 내지 제 42항 중 어느 한 항에 있어서,
상기 좌표들 중 하나 이상에 대해, 상기 발현값의 조합은 하나 이상의 상기 발현값들에 상응하는 하나 이상의 비선형항을 포함하는 것인, 데이터세트를 분석하는 방법. - 제 39항 내지 제 42항 중 어느 한 항에 있어서,
상기 분석의 결과를 생성하는 단계를 더 포함하는, 데이터세트를 분석하는 방법. - 제 39항 내지 제 44항 중 어느 한 항에 있어서,
상기 데이터세트는 하나 이상의 다차원 항목을 포함하는, 데이터세트를 분석하는 방법. - 제 45항에 있어서,
상기 데이터세트의 각 항목은 각 대상체의 하나 이상의 임상적 파라미터를 포함하는, 데이터세트를 분석하는 방법. - 제 46항에 있어서,
상기 임상적 파라미터는 성별, 연령, 온도, 발병기간 및 체중으로 이루어진 군으로부터 선택되는 것인, 데이터세트를 분석하는 방법. - 제 39항 내지 제 47항 중 어느 한 항에 있어서,
상기 분석은 머신 러닝(machine learning)을 포함하는, 데이터세트를 분석하는 방법. - 제 48항에 있어서,
상기 머신 러닝은 지도되는 머신 러닝을 포함하는, 데이터세트를 분석하는 방법. - 제 48항 또는 제 49 항에 있어서,
상기 머신 러닝은 클러스터링(clustering), 서포트 벡터 머신(support vector machine), 선형 모델링(linear modeling), k-니어리스트 네이버 분석(k-nearest neighbors analysis), 결정 트리 러닝(decision tree learning), 앙상블 학습 절차(ensemble learning procedure), 신경 네트워크(neural networks), 확률 모델(probabilistic model), 그래픽 모델(graphical model), 베이지안 네트워크(Bayesian network) 로지스틱 회귀(logistic regression) 및 협회 규칙 학습(association rule learning)으로 이루어진 군으로부터 선택되는 하나 이상의 절차를 포함하는, 데이터세트를 분석하는 방법. - 제 50항에 있어서,
상기 머신 러닝은 서포트 벡터 머신, 신경 네트워크 및 로지스틱 회귀로 이루어진 군으로부터 선택되는 것인, 데이터세트를 분석하는 방법. - 제 39항 내지 제 51항 중 어느 한 항에 있어서,
상기 혈액 샘플은 전혈인, 데이터세트를 분석하는 방법. - 제 39항 내지 제 51항 중 어느 한 항에 있어서,
상기 혈액 샘플은 전혈의 분획인, 데이터세트를 분석하는 방법. - 제 53항에 있어서,
상기 전혈의 분획은 혈청 또는 혈장을 포함하는, 데이터세트를 분석하는 방법. - 제 39항 내지 제 54항 중 어느 한 항에 있어서,
상기 발현값은 전기영동적으로 또는 면역화학적으로 결정되는 것인, 데이터세트를 분석하는 방법. - 제 55항에 있어서,
상기 면역화학적 결정은 유세포 분석법(flow cytometry), 방사 면역 분석법(radioimmunoassay), 면역 형광법(immunofluorescence) 또는 효소 결합 면역 흡착 분석법(enzyme-linked immunosorbent assay)에 의해 수행되는 것인, 데이터세트를 분석하는 방법. - 질병을 가진 대상체 내 TRAIL 단백질 혈청 레벨을 계산하는 단계를 포함하는 질병의 예후를 예측하는 방법으로,
상기 TRAIL 단백질 혈청 레벨이 미리 결정된 레벨 이하인 경우, 미리 결정된 레벨 이상의 TRAIL 단백질 혈청 레벨의 질병을 가진 대상체보다 예후가 악화된 것인, 질병의 예후를 예측하는 방법. - 제 57항에 있어서,
상기 질병은 감염성 질병인 것인, 질병의 예후를 예측하는 방법. - 제 57항에 있어서,
상기 질병은 감염성 질병이 아닌, 질병의 예후를 예측하는 방법. - 제 57항에 있어서,
상기 미리 결정된 레벨은 20 pg/ml 이하인, 질병의 예후를 예측하는 방법. - 대상체 내 TRAIL 단백질 혈청 레벨을 계산하는 것을 포함하는, 대상체의 질병 치료 과정을 결정하는 방법으로,
상기 TRAIL 레벨이 미리 결정된 레벨 이하인 경우, 상기 대상체는 최후의 수단으로 치료받게 되는 것인, 질병 치료 과정을 결정하는 방법. - 제 61항에 있어서,
상기 미리 결정된 레벨은 20 pg/ml 이하인, 질병 치료 과정을 결정하는 방법. - 대상체의 TRAIL 단백질 혈청 레벨을 임신 가능한 건강한 여성 또는 임신 가능한 건강한 여성 대상체 그룹의 TRAIL 단백질 혈청 레벨에 상응하는 미리 결정된 임계값과 비교하는 단계를 포함하는 임신 가능한 여성 대상체에서 감염 유형을 결정하는 방법으로, 상기 TRAIL 단백질 혈청 레벨과 상기 미리 결정된 임계값 간의 차이가 감염 유형을 나타내는 것인, 감염 유형을 결정하는 방법.
- 대상체의 TRAIL 단백질 혈청 레벨을 임신 가능한 건강한 남성 또는 임신 가능한 건강한 남성 대상체 그룹의 TRAIL 단백질 혈청 레벨에 상응하는 미리 결정된 임계값과 비교하는 단계를 포함하는 임신 가능한 남성 대상체에서 감염 유형을 결정하는 방법으로, 상기 TRAIL 단백질 혈청 레벨과 상기 미리 결정된 임계값 간의 차이가 감염 유형을 나타내는 것인, 감염 유형을 결정하는 방법.
- 제 63항 또는 제64항에 있어서,
상기 TRAIL 단백질 혈청 레벨이 미리 결정된 레벨 이상인 경우, 감염 유형은 바이러스성인, 감염 유형을 결정하는 방법. - 제 63항 또는 제 64항에 있어서,
상기 TRAIL 단백질 혈청 레벨이 미리 결정된 레벨 이상인 경우, 감염 유형은 박테리아성이 아닌, 감염 유형을 결정하는 방법. - 제 63항 또는 제 64항에 있어서,
상기 TRAIL 단백질 혈청 레벨이 미리 결정된 레벨 이하인 경우, 감염 유형은 박테리아성인, 감염 유형을 결정하는 방법. - 제 63항 또는 제 64항에 있어서,
상기 TRAIL 단백질 혈청 레벨이 미리 결정된 레벨 이하인 경우, 감염 유형은 바이러스성이 아닌, 감염 유형을 결정하는 방법. - 계산 활성화 제어를 갖는 그래픽 사용자 인터페이스(GUI)를 디스플레이 장치 상에 디스플레이하는 단계;
대상체의 혈액 내에서 폴리펩타이드의 발현값을 얻는 단계;
사용자에 의한 상기 활성화 제어에 응답하여 상기 발현값에 기초하여 점수를 자동적으로 계산하는 단계;
상기 대상체의 박테리아성 감염에 상응하는 것으로 식별된 제 1 단부 및 상기 대상체의 바이러스성 감염에 상응하는 것으로 식별된 제 2 단부 갖는 그래픽 스케일을 GUI 상에 생성하는 단계; 및
상기 점수에 상응하는 위치에서 상기 스케일 상에 마크를 생성하는 단계;를 포함하는, 생물학적 데이터를 분석하기 위해 컴퓨터 구현 방법. - 제 69항에 있어서,
상기 발현값을 얻는 단계는 상기 발현값을 계산하는 외부 머신과 통신하는 단계를 포함하는, 생물학적 데이터를 분석하기 위해 컴퓨터 구현 방법. - 제 70항에 있어서,
상기 GUI는 통신 제어를 더 포함하고, 상기 외부 머신과 통신하는 단계는 유저에 의한 통신 제어 활성에 반응하는 것인, 생물학적 데이터를 분석하기 위해 컴퓨터 구현 방법. - 제 69항에 있어서,
상기 GUI는 복수의 발현값 입력 필드를 포함하고, 상기 발현값을 얻는 단계는 상기 입력 필드에 의한 것인, 생물학적 데이터를 분석하기 위해 컴퓨터 구현 방법. - 제 69항 내지 제 72항 중 어느 한 항에 있어서,
상기 점수는 상기 대상체의 박테리아성 감염 가능성을 나타내는 것인, 생물학적 데이터를 분석하기 위해 컴퓨터 구현 방법. - 제 69항 내지 제 73항 중 어느 한 항에 있어서,
상기 점수는 상기 대상체의 바이러스성 감염 가능성을 나타내는 것인, 생물학적 데이터를 분석하기 위해 컴퓨터 구현 방법.
Applications Claiming Priority (5)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201462037180P | 2014-08-14 | 2014-08-14 | |
| US62/037,180 | 2014-08-14 | ||
| US201562105938P | 2015-01-21 | 2015-01-21 | |
| US62/105,938 | 2015-01-21 | ||
| PCT/IL2015/050823 WO2016024278A1 (en) | 2014-08-14 | 2015-08-12 | Computational analysis of biological data using manifold and a hyperplane |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20170041907A true KR20170041907A (ko) | 2017-04-17 |
Family
ID=55303939
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020177007002A KR20170041907A (ko) | 2014-08-14 | 2015-08-12 | 매니폴드 및 초평면을 이용한 생물학적 데이터의 컴퓨터 분석법 |
Country Status (12)
| Country | Link |
|---|---|
| US (4) | US10303846B2 (ko) |
| EP (2) | EP3180621B1 (ko) |
| JP (2) | JP6661607B2 (ko) |
| KR (1) | KR20170041907A (ko) |
| CN (2) | CN107076746B (ko) |
| AU (2) | AU2015302870B2 (ko) |
| BR (1) | BR112017002884A2 (ko) |
| CA (2) | CA2954601C (ko) |
| IL (1) | IL250585B (ko) |
| RU (1) | RU2730836C2 (ko) |
| WO (1) | WO2016024278A1 (ko) |
| ZA (1) | ZA201700939B (ko) |
Families Citing this family (35)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN106442984B (zh) | 2010-04-21 | 2020-03-13 | 米密德诊断学有限公司 | 区分细菌与病毒感染的标记物和决定因素以及其使用方法 |
| CA2863819C (en) | 2012-02-09 | 2021-11-23 | Memed Diagnostics Ltd. | Signatures and determinants for diagnosing infections and methods of use thereof |
| EP3180621B1 (en) | 2014-08-14 | 2020-04-01 | Memed Diagnostics Ltd. | Computational analysis of biological data using manifold and a hyperplane |
| WO2016059636A1 (en) | 2014-10-14 | 2016-04-21 | Memed Diagnostics Ltd. | Signatures and determinants for diagnosing infections in non-human subjects and methods of use thereof |
| US11868851B2 (en) * | 2015-03-11 | 2024-01-09 | Symphonyai Sensa Llc | Systems and methods for predicting outcomes using a prediction learning model |
| US20190050793A1 (en) * | 2016-02-12 | 2019-02-14 | Carrier Corporation | Shelf life estimation system |
| CN108699583B (zh) | 2016-03-03 | 2022-11-01 | 米密德诊断学有限公司 | 用于区分细菌和病毒感染的rna决定子 |
| US20200124593A1 (en) * | 2016-06-23 | 2020-04-23 | Memed Diagnostics Ltd. | Measuring trail by lateral flow immunoassay |
| CA3027341A1 (en) | 2016-07-10 | 2018-01-18 | Memed Diagnostics Ltd. | Protein signatures for distinguishing between bacterial and viral infections |
| US11340223B2 (en) | 2016-07-10 | 2022-05-24 | Memed Diagnostics Ltd. | Early diagnosis of infections |
| CN109844532B (zh) * | 2016-08-10 | 2022-06-17 | 米密德诊断学有限公司 | 分析生物数据的系统和方法 |
| KR101739703B1 (ko) * | 2016-09-13 | 2017-05-24 | (주) 어드밴스드 엔티 | Trail 기반 뇌염 감별진단 방법 및 장치 |
| US11353456B2 (en) | 2016-09-29 | 2022-06-07 | Memed Diagnostics Ltd. | Methods of risk assessment and disease classification for appendicitis |
| US11385241B2 (en) | 2016-09-29 | 2022-07-12 | Memed Diagnostics Ltd. | Methods of prognosis and treatment |
| EP3568864A1 (en) | 2017-01-11 | 2019-11-20 | Koninklijke Philips N.V. | Method and system for automated inclusion or exclusion criteria detection |
| WO2018152527A1 (en) * | 2017-02-17 | 2018-08-23 | Accuweather, Inc. | System and method for forecasting economic trends using statistical analysis of weather data |
| US20200035214A1 (en) * | 2017-03-16 | 2020-01-30 | Mitsubishi Electric Corporation | Signal processing device |
| US10209260B2 (en) | 2017-07-05 | 2019-02-19 | Memed Diagnostics Ltd. | Signatures and determinants for diagnosing infections and methods of use thereof |
| US10474681B1 (en) * | 2017-09-26 | 2019-11-12 | Microsoft Technology Licensing, Llc | Filtering mapped datasets |
| US11504071B2 (en) * | 2018-04-10 | 2022-11-22 | Hill-Rom Services, Inc. | Patient risk assessment based on data from multiple sources in a healthcare facility |
| US11908581B2 (en) | 2018-04-10 | 2024-02-20 | Hill-Rom Services, Inc. | Patient risk assessment based on data from multiple sources in a healthcare facility |
| WO2020036571A1 (en) | 2018-08-16 | 2020-02-20 | RICHARDSON, Paul, Stephen | Systems and methods for automatic bias monitoring of cohort models and un-deployment of biased models |
| TW202018727A (zh) | 2018-11-09 | 2020-05-16 | 財團法人工業技術研究院 | 整體式學習預測方法與系統 |
| FR3088652A1 (fr) * | 2018-11-21 | 2020-05-22 | Nanotracks Diagnostics | Détection in vitro de la présence d’au moins un agent pathogène microbien dans un échantillon biologique |
| WO2020169511A1 (en) * | 2019-02-18 | 2020-08-27 | The University Of Liverpool | Method of diagnosis |
| CA3132655A1 (en) * | 2019-03-12 | 2020-09-17 | Novartis Ag | Digital solutions for differentiating asthma from copd |
| US11104946B2 (en) * | 2019-04-23 | 2021-08-31 | Inflammatix, Inc. | DNA sequences related to diagnosis and treatment of systemic inflammatory response syndrome |
| CN111816297A (zh) * | 2020-03-31 | 2020-10-23 | 复旦大学附属中山医院 | 一种基于云端的nCoV病毒诊疗系统和方法 |
| EP4152000A4 (en) * | 2020-06-30 | 2024-07-03 | Horiba Ltd | BODY FLUID ANALYSIS DEVICE, BODY FLUID SAMPLE DETERMINATION METHOD AND COMPUTER PROGRAM |
| CN111657961B (zh) * | 2020-07-03 | 2021-04-27 | 北京印刷学院 | 一种在皮肤上制备超表面用于血糖检测的方法 |
| JP2023145811A (ja) * | 2020-08-17 | 2023-10-12 | 孝章 赤池 | 学習モデルの生成方法、プログラム、演算装置 |
| CN112185583B (zh) * | 2020-10-14 | 2022-05-31 | 天津之以科技有限公司 | 一种基于贝叶斯网络的数据挖掘检疫方法 |
| US11430575B2 (en) * | 2020-11-13 | 2022-08-30 | Specialty Diagnostic (SDI) Laboratories, Inc. | Systems and methods for a data driven disease test result prediction |
| CN112632151B (zh) * | 2020-12-25 | 2023-02-10 | 南京邮电大学 | 一种基于二分过滤的移动对象传染行为挖掘方法 |
| US11361847B1 (en) | 2021-02-06 | 2022-06-14 | Timothy A. Hodge | System and method for rapidly reporting testing results |
Family Cites Families (159)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| NL154600B (nl) | 1971-02-10 | 1977-09-15 | Organon Nv | Werkwijze voor het aantonen en bepalen van specifiek bindende eiwitten en hun corresponderende bindbare stoffen. |
| NL154598B (nl) | 1970-11-10 | 1977-09-15 | Organon Nv | Werkwijze voor het aantonen en bepalen van laagmoleculire verbindingen en van eiwitten die deze verbindingen specifiek kunnen binden, alsmede testverpakking. |
| NL154599B (nl) | 1970-12-28 | 1977-09-15 | Organon Nv | Werkwijze voor het aantonen en bepalen van specifiek bindende eiwitten en hun corresponderende bindbare stoffen, alsmede testverpakking. |
| US3901654A (en) | 1971-06-21 | 1975-08-26 | Biological Developments | Receptor assays of biologically active compounds employing biologically specific receptors |
| US3853987A (en) | 1971-09-01 | 1974-12-10 | W Dreyer | Immunological reagent and radioimmuno assay |
| US3867517A (en) | 1971-12-21 | 1975-02-18 | Abbott Lab | Direct radioimmunoassay for antigens and their antibodies |
| NL171930C (nl) | 1972-05-11 | 1983-06-01 | Akzo Nv | Werkwijze voor het aantonen en bepalen van haptenen, alsmede testverpakkingen. |
| US3850578A (en) | 1973-03-12 | 1974-11-26 | H Mcconnell | Process for assaying for biologically active molecules |
| US3935074A (en) | 1973-12-17 | 1976-01-27 | Syva Company | Antibody steric hindrance immunoassay with two antibodies |
| US3996345A (en) | 1974-08-12 | 1976-12-07 | Syva Company | Fluorescence quenching with immunological pairs in immunoassays |
| US4034074A (en) | 1974-09-19 | 1977-07-05 | The Board Of Trustees Of Leland Stanford Junior University | Universal reagent 2-site immunoradiometric assay using labelled anti (IgG) |
| US3984533A (en) | 1975-11-13 | 1976-10-05 | General Electric Company | Electrophoretic method of detecting antigen-antibody reaction |
| US4098876A (en) | 1976-10-26 | 1978-07-04 | Corning Glass Works | Reverse sandwich immunoassay |
| CA1178414A (en) | 1978-02-08 | 1984-11-27 | Toyo Boseki Kabushiki Kaisha (Trading Under The Name Of Toyobo Co., Ltd.) | Packaging material having excellent seal packaging property |
| US4275149A (en) | 1978-11-24 | 1981-06-23 | Syva Company | Macromolecular environment control in specific receptor assays |
| US4233402A (en) | 1978-04-05 | 1980-11-11 | Syva Company | Reagents and method employing channeling |
| US4376110A (en) | 1980-08-04 | 1983-03-08 | Hybritech, Incorporated | Immunometric assays using monoclonal antibodies |
| US4879219A (en) | 1980-09-19 | 1989-11-07 | General Hospital Corporation | Immunoassay utilizing monoclonal high affinity IgM antibodies |
| US4659678A (en) | 1982-09-29 | 1987-04-21 | Serono Diagnostics Limited | Immunoassay of antigens |
| US4727022A (en) | 1984-03-14 | 1988-02-23 | Syntex (U.S.A.) Inc. | Methods for modulating ligand-receptor interactions and their application |
| US5011771A (en) | 1984-04-12 | 1991-04-30 | The General Hospital Corporation | Multiepitopic immunometric assay |
| US4666828A (en) | 1984-08-15 | 1987-05-19 | The General Hospital Corporation | Test for Huntington's disease |
| US4683202A (en) | 1985-03-28 | 1987-07-28 | Cetus Corporation | Process for amplifying nucleic acid sequences |
| US4801531A (en) | 1985-04-17 | 1989-01-31 | Biotechnology Research Partners, Ltd. | Apo AI/CIII genomic polymorphisms predictive of atherosclerosis |
| US5018067A (en) | 1987-01-12 | 1991-05-21 | Iameter Incorporated | Apparatus and method for improved estimation of health resource consumption through use of diagnostic and/or procedure grouping and severity of illness indicators |
| US5272057A (en) | 1988-10-14 | 1993-12-21 | Georgetown University | Method of detecting a predisposition to cancer by the use of restriction fragment length polymorphism of the gene for human poly (ADP-ribose) polymerase |
| US5192659A (en) | 1989-08-25 | 1993-03-09 | Genetype Ag | Intron sequence analysis method for detection of adjacent and remote locus alleles as haplotypes |
| DE4322330A1 (de) | 1992-08-31 | 1994-03-03 | Behringwerke Ag | Verwendung des IL-4-Rezeptors zur Therapie, Prophylaxe und Diagnose von allergischen, viralen, parasitären und bakteriellen Erkrankungen sowie von Pilzinfektionen |
| US5281521A (en) | 1992-07-20 | 1994-01-25 | The Trustees Of The University Of Pennsylvania | Modified avidin-biotin technique |
| DE4227454C1 (de) | 1992-08-19 | 1994-02-03 | Henning Berlin Gmbh | Verfahren zur Früherkennung, zur Erkennung des Schweregrads sowie zur therapiebegleitenden Verlaufsbeurteilung einer Sepsis sowie Mittel zur Durchführung des Verfahrens |
| US6283761B1 (en) | 1992-09-08 | 2001-09-04 | Raymond Anthony Joao | Apparatus and method for processing and/or for providing healthcare information and/or healthcare-related information |
| SE9401351D0 (sv) | 1994-04-21 | 1994-04-21 | Venge | A method for diagnosis |
| US5910421A (en) | 1995-12-21 | 1999-06-08 | University Of Florida | Rapid diagnostic method for distinguishing allergies and infections |
| US6077665A (en) | 1996-05-07 | 2000-06-20 | The Board Of Trustees Of The Leland Stanford Junior University | Rapid assay for infection in neonates |
| EP1042459A4 (en) | 1997-12-24 | 2003-07-23 | Diatech Pty Ltd | BIFUNCTIONAL MOLECULES |
| CA2329868A1 (en) | 1998-05-18 | 1999-11-25 | Apoptosis Technology, Inc. | Compounds, screening methods, and uses involving anti-apoptotic genes and gene products |
| DE19847690A1 (de) | 1998-10-15 | 2000-04-20 | Brahms Diagnostica Gmbh | Verfahren und Substanzen für die Diagnose und Therapie von Sepsis und sepsisähnlichen systemischen Infektionen |
| US6709855B1 (en) | 1998-12-18 | 2004-03-23 | Scios, Inc. | Methods for detection and use of differentially expressed genes in disease states |
| WO2000040749A2 (en) | 1999-01-06 | 2000-07-13 | Genenews Inc. | Method for the detection of gene transcripts in blood and uses thereof |
| CA2384978C (en) | 1999-08-24 | 2012-10-23 | Glycozym Aps | Udp-n-acetylglucosamine: galactose-.beta.1,3-n-acetylgalactosamine-.alpha.-r/(glcnac to galnac) .beta.1,6-n-acetylglucosaminyltransferase, c2gnt3 |
| IL132687A0 (en) * | 1999-11-01 | 2001-03-19 | Keren Mechkarim Ichilov Pnimit | System and method for evaluating body fluid samples |
| US20020038227A1 (en) | 2000-02-25 | 2002-03-28 | Fey Christopher T. | Method for centralized health data management |
| US7668661B2 (en) * | 2000-04-28 | 2010-02-23 | Siemens Healthcare Diagnostics Inc. | Liver disease-related methods and systems |
| RU2229719C2 (ru) | 2001-03-27 | 2004-05-27 | Кабусики Кайся Тосиба | Обнаружение имеющей отношение к заболеванию нуклеиновой кислоты |
| US7713705B2 (en) * | 2002-12-24 | 2010-05-11 | Biosite, Inc. | Markers for differential diagnosis and methods of use thereof |
| DE10119804A1 (de) | 2001-04-23 | 2002-10-24 | Brahms Ag | Entzündungsspezifische Peptide und deren Verwendungen |
| JP3665316B2 (ja) * | 2001-05-18 | 2005-06-29 | 麒麟麦酒株式会社 | 抗trail−r抗体 |
| FI115165B (fi) | 2001-06-04 | 2005-03-15 | Aboatech Ab Oy | Menetelmä infektion laadun määrittämiseksi |
| DE10130985B4 (de) | 2001-06-27 | 2004-03-18 | B.R.A.H.M.S Ag | Verfahren zur Diagnose von Sepsis und schweren Infektionen unter Bestimmung löslicher Cytokeratin-1-Fragmente |
| US20040209307A1 (en) | 2001-08-20 | 2004-10-21 | Biosite Incorporated | Diagnostic markers of stroke and cerebral injury and methods of use thereof |
| EP1318407B1 (de) | 2001-12-07 | 2004-09-29 | B.R.A.H.M.S Aktiengesellschaft | Verwendungen der Aldose-1-Epimerase (Mutarotase) für die Diagnose von Entzündungserkrankungen und Sepsis |
| EP1317902B1 (en) | 2001-12-10 | 2005-11-09 | Kabushiki Gaisha K-and-S | Biological data observation apparatus |
| US20040038201A1 (en) | 2002-01-22 | 2004-02-26 | Whitehead Institute For Biomedical Research | Diagnostic and therapeutic applications for biomarkers of infection |
| US7468032B2 (en) | 2002-12-18 | 2008-12-23 | Cardiac Pacemakers, Inc. | Advanced patient management for identifying, displaying and assisting with correlating health-related data |
| US20040122296A1 (en) | 2002-12-18 | 2004-06-24 | John Hatlestad | Advanced patient management for triaging health-related data |
| US20050227223A1 (en) | 2002-03-22 | 2005-10-13 | Toshio Miyawaki | Method of judging viral infection |
| US7485298B2 (en) | 2002-05-23 | 2009-02-03 | Michael Powell | Diagnosis and treatment of human dormancy-related sequellae |
| KR20050040866A (ko) | 2002-06-11 | 2005-05-03 | 아이다호 리서치 파운데이션 | 바이러스 감염의 검출을 위한 ⅰ형 인터페론-유도성 단백질 |
| US8563476B2 (en) | 2002-11-15 | 2013-10-22 | Morehouse School Of Medicine | Anti-CXCL9, anti-CXCL10, anti-CXCL11, anti-CXCL13, anti-CXCR3 and anti-CXCR5 agents for inflammatory disorders |
| WO2004108899A2 (en) | 2003-06-04 | 2004-12-16 | The Government Of The United States As Represented By The Secretary Of The Department Of Health And Human Services, Centers For Disease Control And Prevention | Pni microarray and uses |
| JP2007518062A (ja) | 2003-09-29 | 2007-07-05 | バイオサイト インコーポレイテッド | 敗血症を診断する方法および診断するための組成物 |
| JP2005106694A (ja) * | 2003-09-30 | 2005-04-21 | Mochida Pharmaceut Co Ltd | 敗血症早期検出及び重篤度評価 |
| GB0401730D0 (en) | 2004-01-27 | 2004-03-03 | Bioxell Spa | Diagnosis method |
| US8906630B2 (en) | 2004-02-27 | 2014-12-09 | The Trustees Of The University Of Pennsylvania | Assays for detecting pathogenic respiratory bacteria |
| US7892539B2 (en) | 2004-03-24 | 2011-02-22 | The Regents Of The University Of California | Modulation of an innate immune response by altering TRIAL-R signaling |
| US8155993B2 (en) * | 2004-06-15 | 2012-04-10 | Janssen Pharmaceutica, N.V. | Apparatus and methods for assessing a pharmaceutical product |
| AU2005327199A1 (en) * | 2004-07-09 | 2006-08-17 | Amaox, Inc. | Immune cell biosensors and methods of using same |
| US7572640B2 (en) | 2004-09-28 | 2009-08-11 | Singulex, Inc. | Method for highly sensitive detection of single protein molecules labeled with fluorescent moieties |
| AU2005290314A1 (en) * | 2004-09-28 | 2006-04-06 | Singulex, Inc. | System and method for spectroscopic analysis of single particles |
| EP1807540A4 (en) | 2004-11-05 | 2008-12-10 | Us Gov Sec Navy | DIAGNOSIS AND PROGNOSIS OF CLINICAL PHENOTYPES OF INFECTIOUS DISEASES AND OTHER BIOLOGICAL CONDITIONS USING HOST GENE EXPRESSION MARKERS IN BLOOD |
| US20060099628A1 (en) | 2004-11-10 | 2006-05-11 | Wei-Mei Ching | Diagnostic assay for rickettsia prowazekii disease by detection of responsive gene expression |
| CN101208602A (zh) | 2005-04-15 | 2008-06-25 | 贝克顿迪金森公司 | 脓毒症的诊断 |
| KR20080006617A (ko) | 2005-04-15 | 2008-01-16 | 백톤 디킨슨 앤드 컴퍼니 | 패혈증의 진단 |
| US20070015172A1 (en) | 2005-06-01 | 2007-01-18 | Z-Biomed, Inc. | Expression profiles for microbial infection |
| UA78641C2 (en) | 2005-08-29 | 2007-04-10 | Open Joint Stock Company Conce | A process for the preparation of carbamide-formaldehyde resin |
| DE102005050933A1 (de) | 2005-10-21 | 2007-04-26 | Justus-Liebig-Universität Giessen | Erfindung betreffend Expressionsprofile zur Vorhersage von septischen Zuständen |
| CN101378764A (zh) | 2005-12-09 | 2009-03-04 | 贝勒研究院 | 通过血液白细胞微阵列分析对系统性红斑狼疮的诊断、预后和疾病发展的监测 |
| GB0601959D0 (en) | 2006-01-31 | 2006-03-15 | King S College London | Sepsis test |
| US20070184460A1 (en) | 2006-02-09 | 2007-08-09 | Wei-Mei Ching | Diagnostic assay for Orientia tsutsugamushi by detection of responsive gene expression |
| EP2016195A4 (en) | 2006-04-26 | 2010-03-10 | Vertex Pharma | BIOMARKERS FOR HEPATITIS C VIRUS INFECTION |
| RU2348695C2 (ru) * | 2006-05-23 | 2009-03-10 | Закрытое акционерное общество "Молекулярно-медицинские технологии" | Дифференцирующий и специфический олигонуклеотиды для идентификации последовательностей днк инфекционных агентов в биологических материалах, способ видовой идентификации инфекционных агентов, биочип и набор для осуществления этого способа |
| AU2007286915B2 (en) | 2006-08-11 | 2014-05-15 | Baylor Research Institute | Gene expression signatures in blood leukocytes permit differential diagnosis of acute infections |
| ATE504000T1 (de) | 2006-09-05 | 2011-04-15 | Hvidovre Hospital | Auf ip-10 basierende immunologische überwachung |
| AU2012244350B2 (en) | 2006-09-05 | 2013-08-15 | Statens Serum Institut | Ip-10 based immunological monitoring |
| JP2010506166A (ja) | 2006-10-05 | 2010-02-25 | エージェンシー フォー サイエンス,テクノロジー アンド リサーチ | デング熱の診断及び治療 |
| BRPI0720035A2 (pt) | 2006-12-06 | 2019-05-07 | Medimmune, Llc | métodos para tratar um paciente tendo uma doença ou distúrbio, para tratar um paciente com doença autoimune, para neutralizar um perfil de expressão do marcador pd indutível por ifn do tipo 1 ou ifnalfa em um paciente, para monitorar ou prognosticar progressão de doença autoimune de um paciente, para monitorar a progressão da doença de um paciente que recebe o tratamento com um agente terapêutico que liga-se a e modula a atividade de ifnalfa, para identificar um paciente como um candidato para um agente terapêutico que liga-se a e modula a atividade de ifnalfa, para diagnosticar um paciente como tendo um distùrbio associado com níveis aumentados de ifnalfa, para identificar um terapêutico candidato para o tratamento de distùrbios mediados por ifnalfa e para detectar atividade de ifn em uma amostra, conjunto de sondas, e, kit |
| DE102006060112A1 (de) | 2006-12-20 | 2008-06-26 | Brahms Aktiengesellschaft | Diagnose und Risikostratifizierung mittels dem neuen Marker CT-proADM |
| WO2008076077A1 (en) | 2006-12-21 | 2008-06-26 | Innogene Kalbiotech Pte Ltd | Method of early diagnosis of epstein-barr virus-related cancer and respective reagents and kits |
| US20100221700A1 (en) | 2007-01-11 | 2010-09-02 | Haynes Barton F | Method of monitoring hiv infection |
| WO2008095136A2 (en) | 2007-01-31 | 2008-08-07 | Henkin Robert I | Methods for detection of biological substances |
| US20090155180A1 (en) | 2007-02-28 | 2009-06-18 | Mary Lynn Jump | Methods and targets for identifying compounds for regulating rhinovirus infection |
| DE102007009751A1 (de) | 2007-02-28 | 2008-09-04 | B.R.A.H.M.S Aktiengesellschaft | Verfahren zur selektiven Bestimmung von Procalcitonin 1-116 für diagnostische Zwecke sowie Antikörper und Kits zur Durchführung eines solchen Verfahrens |
| US8465951B2 (en) | 2007-06-01 | 2013-06-18 | Council Of Scientific & Industrial Research | Method for simultaneous detection and discrimination of bacterial, fungal, parasitic and viral infections of eye and central nervous system |
| RU2352948C1 (ru) | 2007-06-15 | 2009-04-20 | Государственное образовательное учреждение высшего профессионального образования "КРАСНОЯРСКАЯ ГОСУДАРСТВЕННАЯ МЕДИЦИНСКАЯ АКАДЕМИЯ ФЕДЕРАЛЬНОГО АГЕНТСТВА ПО ЗДРАВООХРАНЕНИЮ И СОЦИАЛЬНОМУ РАЗВИТИЮ" | Способ прогноза исхода заболевания пациентов с ургентной хирургической патологией органов брюшной полости |
| IL184478A (en) | 2007-07-08 | 2017-07-31 | Hadasit Medical Res Services & Development Ltd | Preparations, methods and kits for identifying carriers of mutations in genes 1brca and 2brca and for the early detection of cancers associated with mutations in these genes |
| DE102007035640A1 (de) | 2007-07-27 | 2009-01-29 | Lkh-Kunststoffwerk Gmbh & Co. Kg | Tauchkolben |
| EP2020603A1 (en) | 2007-08-03 | 2009-02-04 | BRAHMS Aktiengesellschaft | Method for risk stratification in stable coronary artery disease |
| ATE533061T1 (de) | 2007-08-03 | 2011-11-15 | Brahms Gmbh | Verfahren zur diagnose bakterieller infektionen |
| TW200920358A (en) | 2007-08-13 | 2009-05-16 | Lundbeck & Co As H | Method of treating stress-mediated depression |
| WO2009025743A2 (en) | 2007-08-17 | 2009-02-26 | University Of Massachusetts Medical School | Use of trail compositions as antiviral agents |
| EP2039980B1 (de) | 2007-09-24 | 2010-04-07 | BMDSys Production GmbH | Kryostat mit stabilisiertem Aussengefäss |
| EP2071334A1 (en) | 2007-12-14 | 2009-06-17 | Transmedi SA | Compositions and methods of detecting TIABS |
| WO2009100907A1 (en) | 2008-02-14 | 2009-08-20 | Dianeering Diagnostics Engineering And Research Gmbh | Means and methods for assessing the risk of patients presenting to emergency departments based on very low concentrations of troponin i or t or using a combination of markers |
| WO2009120800A2 (en) * | 2008-03-25 | 2009-10-01 | University Of Southern California | Prognostic panel for urinary bladder cancer |
| CN102016072A (zh) | 2008-04-21 | 2011-04-13 | 诺瓦提斯研究基金会弗里德里克·米谢尔生物医学研究所 | 抗病毒疗法 |
| US9910036B2 (en) | 2008-05-20 | 2018-03-06 | Rapid Pathogen Screening, Inc. | Method and device for combined detection of viral and bacterial infections |
| CA2721742A1 (en) | 2008-05-23 | 2009-11-26 | Pronota N.V. | New biomarkers for diagnosis, prediction and/or prognosis of sepsis and uses thereof |
| SG182951A1 (en) | 2008-06-25 | 2012-08-30 | Baylor Res Inst | Blood transcriptional signature of mycobacterium tuberculosis infection |
| US20110263821A1 (en) | 2008-10-24 | 2011-10-27 | B.R.A.H.M.S. Gmbh | Prognosis and risk assessment in stroke patients by determining the level of marker peptides |
| TW201021778A (en) | 2008-11-12 | 2010-06-16 | Alcon Res Ltd | Intraocular lens delivery device with a multi-part plunger tip |
| GB2463401B (en) | 2008-11-12 | 2014-01-29 | Caris Life Sciences Luxembourg Holdings S A R L | Characterizing prostate disorders by analysis of microvesicles |
| UA92843C2 (ru) | 2009-05-21 | 2010-12-10 | Институт Технической Теплофизики Национальной Академии Наук Украины | Способ получения порошкообразного пищевого красителя из столовой свеклы |
| WO2010138618A1 (en) | 2009-05-26 | 2010-12-02 | Duke University | Molecular predictors of fungal infection |
| JP5731489B2 (ja) | 2009-06-05 | 2015-06-10 | ベー.エル.アー.ハー.エム.エス ゲゼルシャフト ミット ベシュレンクテル ハフツング | 呼吸困難を起こした患者における細菌感染の検出 |
| US8735080B2 (en) | 2009-08-07 | 2014-05-27 | Rules-Based Medicine, Inc. | Methods and devices for detecting obstructive uropathy and associated disorders |
| JP5667353B2 (ja) * | 2009-09-25 | 2015-02-12 | シスメックス株式会社 | 血球計数装置、診断支援装置、診断支援方法及びコンピュータプログラム |
| ES2601104T3 (es) | 2009-10-13 | 2017-02-14 | B.R.A.H.M.S Gmbh | Procalcitonina para el diagnóstico de infecciones bacterianas y la guía del tratamiento antibiótico en pacientes con apoplejía aguda o accidente isquémico transitorio |
| WO2011047358A1 (en) | 2009-10-15 | 2011-04-21 | Crescendo Bioscience | Biomarkers and methods for measuring and monitoring inflammatory disease activity |
| US20110312534A1 (en) | 2010-03-05 | 2011-12-22 | Erasmus University Medical Center Rotterdam | Method for prediction of human iris color |
| CN106442984B (zh) | 2010-04-21 | 2020-03-13 | 米密德诊断学有限公司 | 区分细菌与病毒感染的标记物和决定因素以及其使用方法 |
| WO2011152012A1 (ja) | 2010-05-31 | 2011-12-08 | 国立大学法人徳島大学 | 病気の重症度の検査方法 |
| GB2482187A (en) * | 2010-07-23 | 2012-01-25 | Univ Exeter | Predicting response to surgery |
| US8877212B2 (en) | 2011-02-07 | 2014-11-04 | Trudeau Institute | Use of an IL12 receptor-beta 1 splice variant to diagnose active tuberculosis |
| RU2476887C2 (ru) | 2011-03-29 | 2013-02-27 | Учреждение Российской академии медицинских наук Дальневосточный научный центр физиологии и патологии дыхания Сибирского отделения РАМН | Способ прогнозирования развития осложнений у больных внебольничной пневмонией |
| AU2012274461A1 (en) * | 2011-06-20 | 2014-01-16 | Kyowa Hakko Kirin Co., Ltd. | Anti-erbB3 antibody |
| US20140329242A1 (en) | 2011-09-12 | 2014-11-06 | Thomas M. Aune | Characterizing multiple sclerosis |
| JP5842107B2 (ja) * | 2011-10-19 | 2016-01-13 | パナソニックIpマネジメント株式会社 | 循環動態測定装置 |
| ES2656150T3 (es) | 2011-11-14 | 2018-02-23 | Universitätsklinikum Jena | Diagnóstico de la sepsis y del síndrome de respuesta inflamatoria sistémica |
| CA2863819C (en) * | 2012-02-09 | 2021-11-23 | Memed Diagnostics Ltd. | Signatures and determinants for diagnosing infections and methods of use thereof |
| EP2637023A1 (en) | 2012-03-08 | 2013-09-11 | B.R.A.H.M.S GmbH | Prediction of outcome in patients with chronic obstructive pulmonary disease |
| WO2013131993A2 (en) | 2012-03-08 | 2013-09-12 | Thommen Medical Ag | Device for the diagnosis of inflammatory tissues in dental applications |
| EP2831264B1 (en) * | 2012-03-30 | 2019-07-10 | VisEn Medical, Inc. | Bacterial imaging agents and methods of using same |
| GB201211982D0 (en) | 2012-07-05 | 2012-08-22 | Isis Innovation | Biomarker |
| WO2014008545A1 (en) | 2012-07-10 | 2014-01-16 | Nepean Blue Mountains Local Health District | Risk stratification in influenza |
| FR2996003B1 (fr) | 2012-09-25 | 2014-10-17 | Commissariat Energie Atomique | Methode pour detecter specifiquement dans un echantillon une metalloprotease matricielle (mmp) d'interet uniquement dans sa forme active |
| US20140127827A1 (en) | 2012-11-05 | 2014-05-08 | Gwangju Institute Of Science And Technology | Method for detection of antigen using fluorescence resonance energy transfer immunoassay |
| CA2893158A1 (en) | 2012-11-30 | 2014-06-05 | Applied Proteomics, Inc. | Method for evaluation of presence of or risk of colon tumors |
| EP3608416A1 (en) | 2013-01-22 | 2020-02-12 | Imicroq, S.L. | Rapid method for detection of pathogen |
| BR112015017954A2 (pt) * | 2013-01-29 | 2017-07-11 | Molecular Health Gmbh | sistemas e métodos para o suporte de decisão clínica |
| US9474903B2 (en) * | 2013-03-15 | 2016-10-25 | Boston Scientific Neuromodulation Corporation | Clinical response data mapping |
| US10689701B2 (en) | 2013-03-15 | 2020-06-23 | Duke University | Biomarkers for the molecular classification of bacterial infection |
| EP3693738A1 (en) | 2013-09-20 | 2020-08-12 | Astute Medical, Inc. | Methods and compositions for diagnosis and prognosis of appendicitis and differentiation of causes of abdominal pain |
| WO2015048098A1 (en) | 2013-09-24 | 2015-04-02 | Washington University | Diagnostic methods for infectious disease using endogenous gene expression |
| EP3180621B1 (en) | 2014-08-14 | 2020-04-01 | Memed Diagnostics Ltd. | Computational analysis of biological data using manifold and a hyperplane |
| WO2016059636A1 (en) | 2014-10-14 | 2016-04-21 | Memed Diagnostics Ltd. | Signatures and determinants for diagnosing infections in non-human subjects and methods of use thereof |
| RU2758608C2 (ru) | 2014-11-19 | 2021-11-01 | Конинклейке Филипс Н.В. | Способ диагностики с использованием hnl |
| CA2968650A1 (en) | 2014-12-11 | 2016-06-16 | Memed Diagnostics Ltd. | Marker combinations for diagnosing infections and methods of use thereof |
| KR20160072626A (ko) | 2014-12-15 | 2016-06-23 | 전북대학교산학협력단 | 패혈증 인자 il-6 간이 진단 키트 |
| EP3367895A4 (en) * | 2015-11-01 | 2019-07-03 | Elminda Ltd. | METHOD AND SYSTEM FOR ESTIMATING POTENTIAL DISTRIBUTION ON A CORTICAL SURFACE |
| CN108699583B (zh) | 2016-03-03 | 2022-11-01 | 米密德诊断学有限公司 | 用于区分细菌和病毒感染的rna决定子 |
| CN114959084A (zh) | 2016-03-03 | 2022-08-30 | 米密德诊断学有限公司 | 分析rna用于诊断感染类型 |
| US20200124593A1 (en) | 2016-06-23 | 2020-04-23 | Memed Diagnostics Ltd. | Measuring trail by lateral flow immunoassay |
| US11340223B2 (en) | 2016-07-10 | 2022-05-24 | Memed Diagnostics Ltd. | Early diagnosis of infections |
| CA3027341A1 (en) | 2016-07-10 | 2018-01-18 | Memed Diagnostics Ltd. | Protein signatures for distinguishing between bacterial and viral infections |
| WO2018052137A1 (ja) | 2016-09-16 | 2018-03-22 | 株式会社オンチップ・バイオテクノロジーズ | 微粒子分注装置、微粒子解析装置、及び反応検出装置、並びにそれらを用いる方法 |
| US11385241B2 (en) | 2016-09-29 | 2022-07-12 | Memed Diagnostics Ltd. | Methods of prognosis and treatment |
| US11353456B2 (en) * | 2016-09-29 | 2022-06-07 | Memed Diagnostics Ltd. | Methods of risk assessment and disease classification for appendicitis |
| US10209260B2 (en) | 2017-07-05 | 2019-02-19 | Memed Diagnostics Ltd. | Signatures and determinants for diagnosing infections and methods of use thereof |
| US20220329345A1 (en) | 2021-04-08 | 2022-10-13 | Infinidome Ltd. | Adjustable null steering in a stationary network |
-
2015
- 2015-08-12 EP EP15831781.8A patent/EP3180621B1/en active Active
- 2015-08-12 RU RU2017107750A patent/RU2730836C2/ru active
- 2015-08-12 KR KR1020177007002A patent/KR20170041907A/ko not_active Application Discontinuation
- 2015-08-12 WO PCT/IL2015/050823 patent/WO2016024278A1/en active Application Filing
- 2015-08-12 BR BR112017002884-0A patent/BR112017002884A2/pt not_active Application Discontinuation
- 2015-08-12 AU AU2015302870A patent/AU2015302870B2/en active Active
- 2015-08-12 US US15/503,439 patent/US10303846B2/en active Active
- 2015-08-12 CN CN201580055946.0A patent/CN107076746B/zh active Active
- 2015-08-12 CN CN202010370187.XA patent/CN111624346A/zh active Pending
- 2015-08-12 JP JP2017507867A patent/JP6661607B2/ja active Active
- 2015-08-12 EP EP20164056.2A patent/EP3699930B1/en active Active
- 2015-08-12 CA CA2954601A patent/CA2954601C/en active Active
- 2015-08-12 CA CA3190715A patent/CA3190715A1/en active Pending
-
2017
- 2017-02-07 ZA ZA2017/00939A patent/ZA201700939B/en unknown
- 2017-02-13 IL IL250585A patent/IL250585B/en active IP Right Grant
-
2019
- 2019-03-18 US US16/355,984 patent/US11081206B2/en active Active
-
2020
- 2020-02-12 JP JP2020021606A patent/JP7095001B2/ja active Active
- 2020-08-20 US US16/998,006 patent/US11450406B2/en active Active
-
2022
- 2022-02-07 AU AU2022200802A patent/AU2022200802A1/en active Pending
- 2022-07-28 US US17/875,467 patent/US11776658B2/en active Active
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| RU2730836C2 (ru) | Вычислительный анализ биологических данных с применением многообразия и гиперплоскости | |
| KR102657306B1 (ko) | 전립선암의 진단 및 치료에서 필라민을 포함하는 마커의 용도 | |
| CN109661578B (zh) | 用于区分细菌和病毒感染的蛋白质特征 | |
| AU2015257483B2 (en) | Biomarkers and combinations thereof for diagnosising tuberculosis | |
| CN101668865B (zh) | 与心血管疾病相关联的遗传易感性变体 | |
| US6117654A (en) | Nucleic acid molecules encoding Tango-77-polypeptides | |
| KR20150090246A (ko) | 암을 위한 분자 진단 테스트 | |
| KR20140044341A (ko) | 암에 대한 분자적 진단 검사 | |
| TW201632629A (zh) | 用於癌症診斷與預後的方法 | |
| KR20110036608A (ko) | 유방암 위험도 평가를 위한 유전적 변이 | |
| CN107478843A (zh) | 用于炎性疾病的抗‑cxcl9、抗‑cxcl10、抗‑cxcl11、抗‑cxcl13、抗‑cxcr3和抗‑cxcr5试剂 | |
| KR102613599B1 (ko) | 뇌경색 발증 리스크 예측 방법 | |
| CA2561669A1 (en) | Methods for identifying risk of osteoarthritis and treatments thereof | |
| TWI849576B (zh) | 基因標記的用途 | |
| CA2497597A1 (en) | Methods for identifying subjects at risk of melanoma and treatments | |
| CA2488876A1 (en) | Activating mutations of platelet derived growth factor receptor alpha (pdgfra) as diagnostic markers and therapeutic targets | |
| KR102326582B1 (ko) | 청각장애의 진단용 마커 및 그의 용도 | |
| US20040138441A1 (en) | Novel gene functionally related to dyslexia | |
| KR20120073261A (ko) | 카텝신 h의 용도 | |
| CN108770360A (zh) | 对癌性疾病进行分期、分型和治疗的手段和方法 | |
| KR102110600B1 (ko) | Frrs1l 유전자 녹아웃 자폐 스펙트럼 장애 동물모델 및 이의 용도 | |
| CN111989572A (zh) | 评估疑似感染、sofa评分低于2的患者中的并发症风险 | |
| KR20230157346A (ko) | 세포외 소포체를 로딩하는 방법 | |
| WO2020260861A1 (en) | Her2 gene splice variants | |
| KR20130048240A (ko) | Pcbp―1 항원에 대한 모노클로날 항체, 및 그의 용도 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A201 | Request for examination | ||
| E902 | Notification of reason for refusal | ||
| E601 | Decision to refuse application |