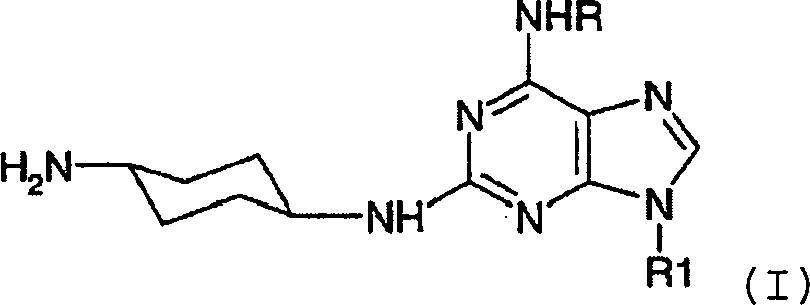
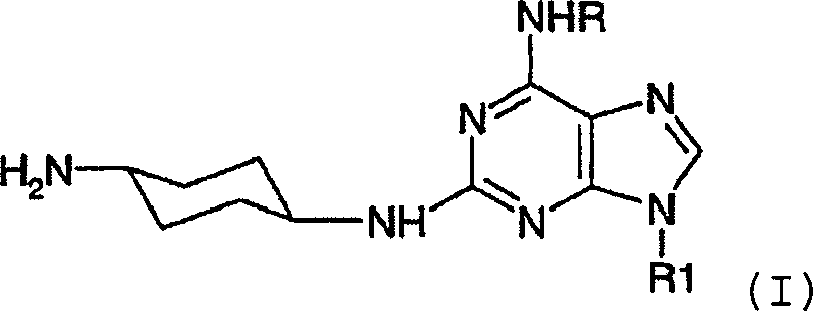
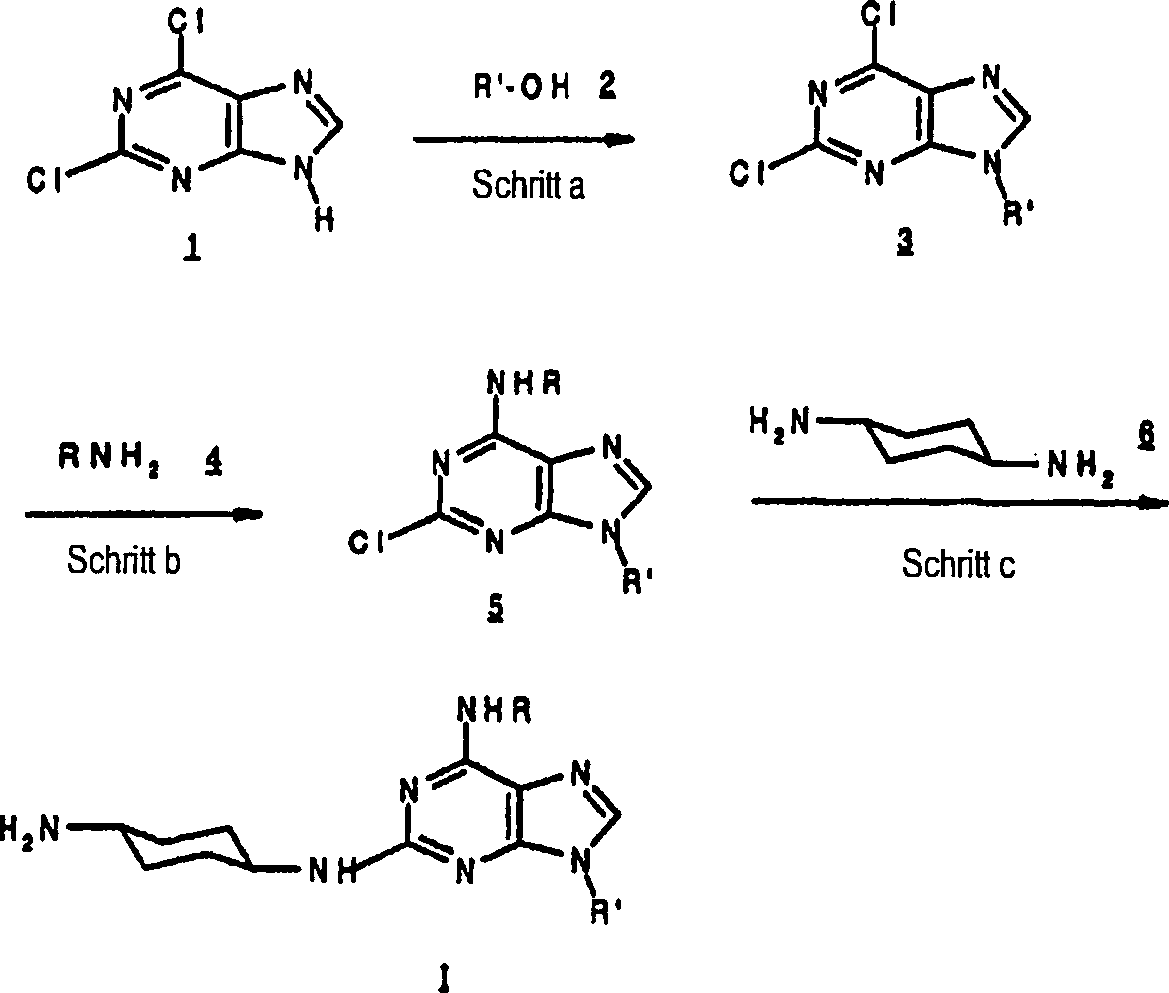
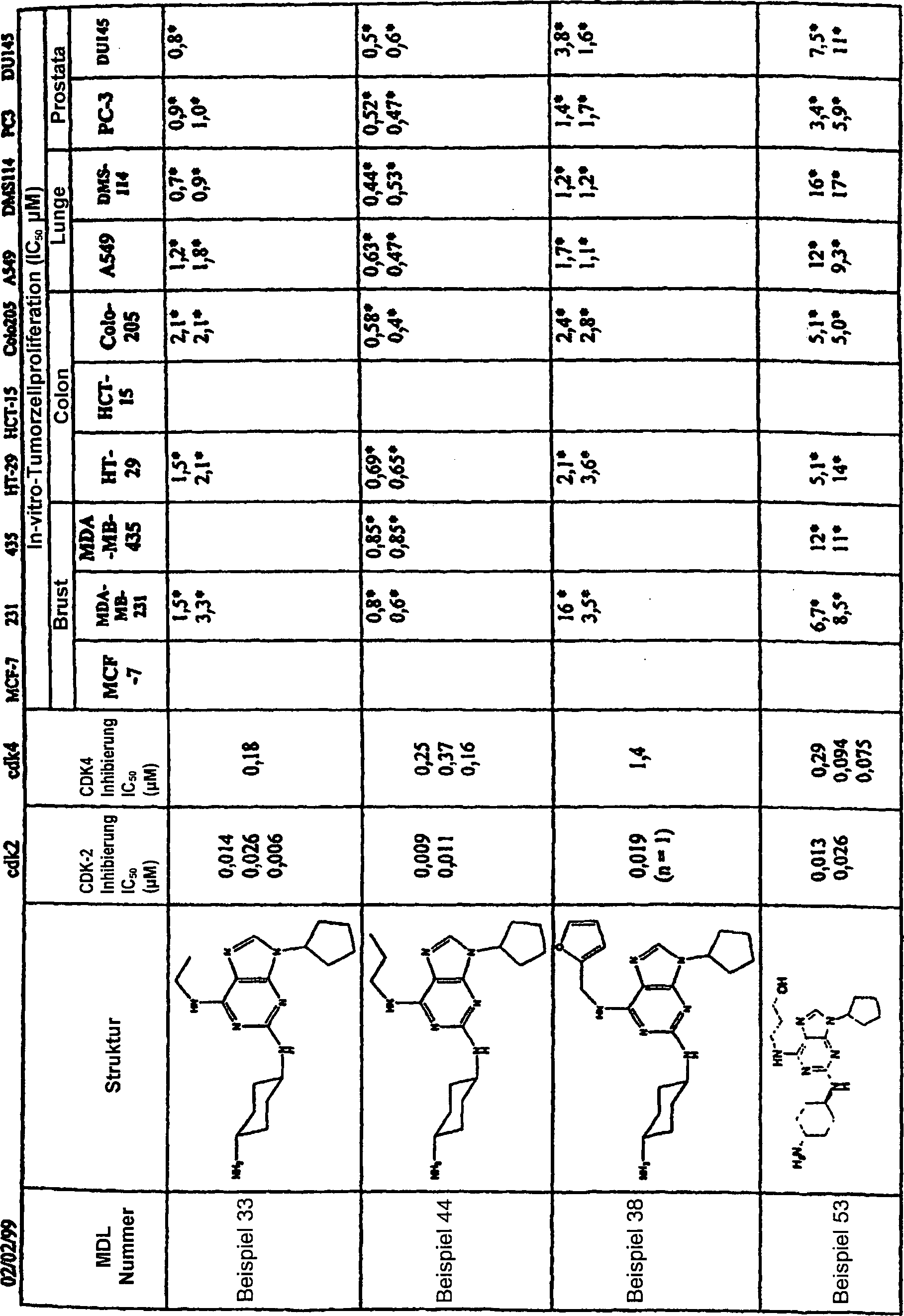
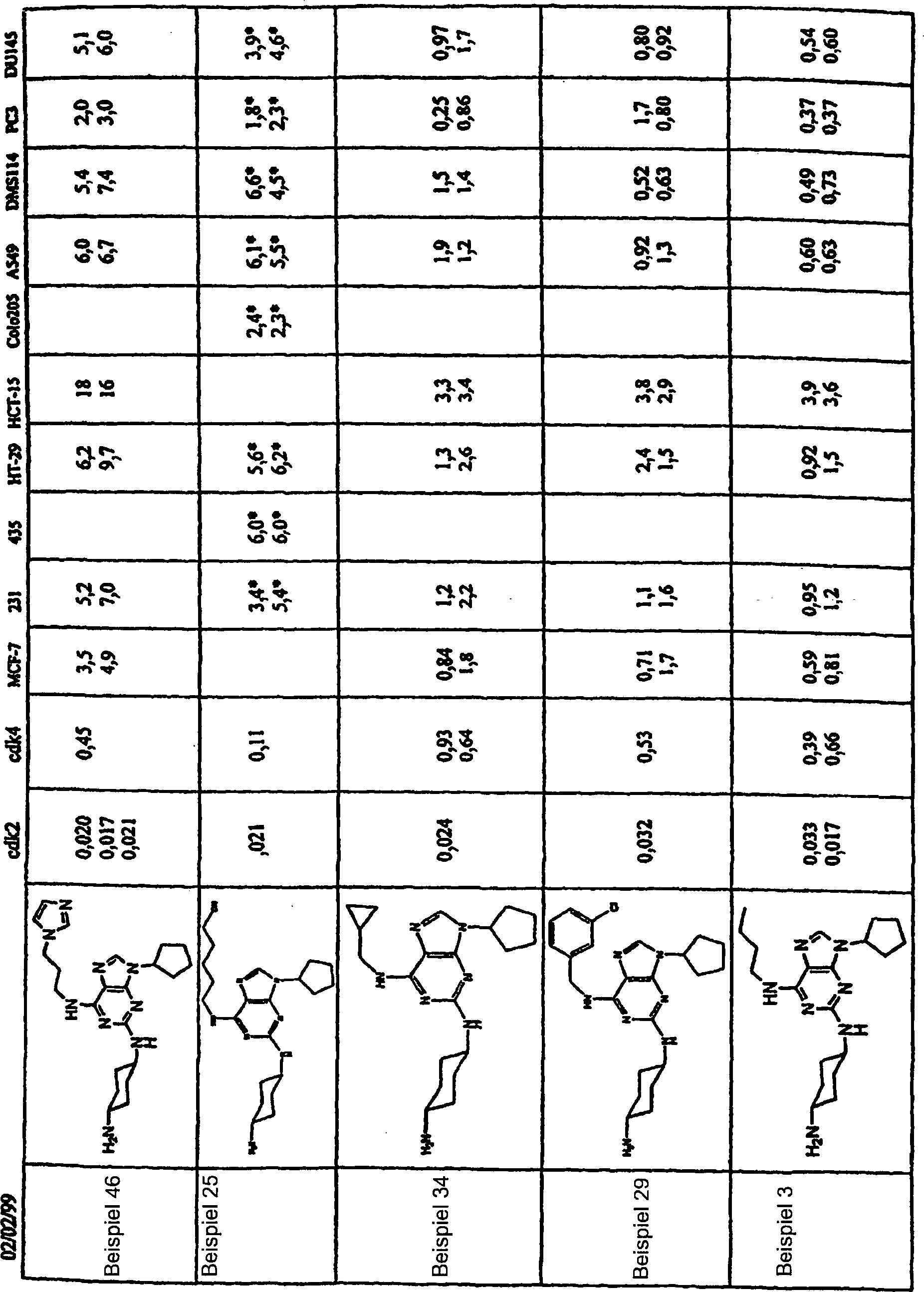
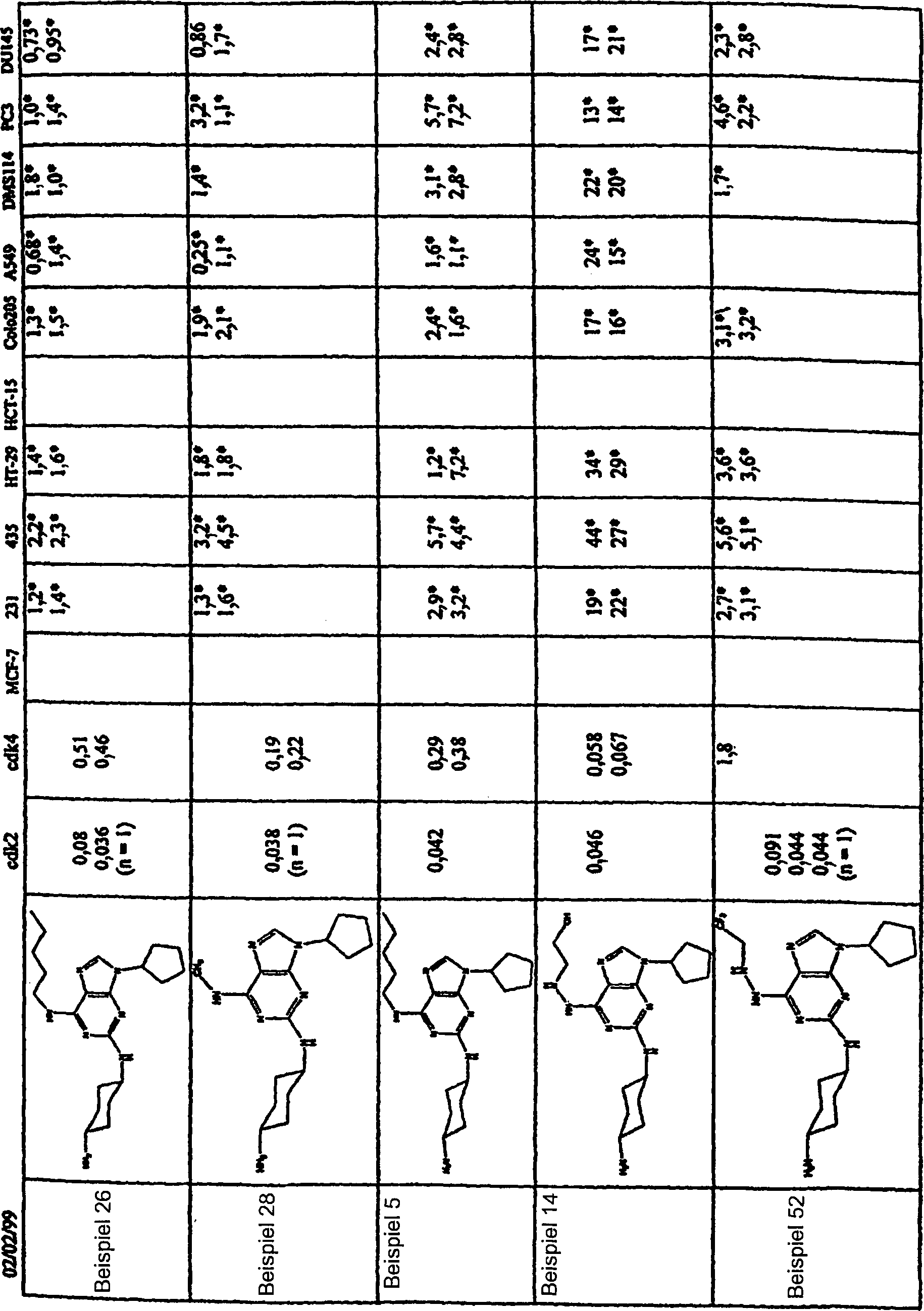
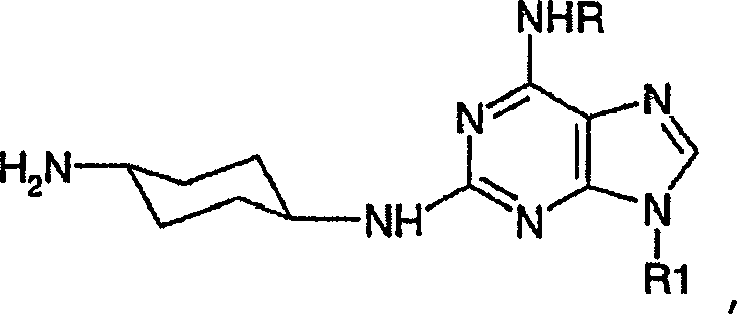
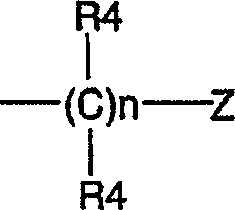
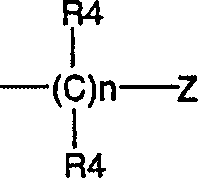
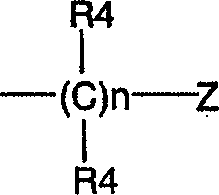
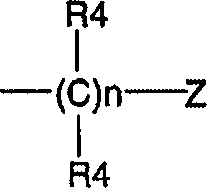
DE69918062T2 - 6,9-disubstituierte 2-(trans-(4-aminocyclohexyl)amino)purine - Google Patents
6,9-disubstituierte 2-(trans-(4-aminocyclohexyl)amino)purine Download PDFInfo
- Publication number
- DE69918062T2 DE69918062T2 DE69918062T DE69918062T DE69918062T2 DE 69918062 T2 DE69918062 T2 DE 69918062T2 DE 69918062 T DE69918062 T DE 69918062T DE 69918062 T DE69918062 T DE 69918062T DE 69918062 T2 DE69918062 T2 DE 69918062T2
- Authority
- DE
- Germany
- Prior art keywords
- amino
- trans
- aminocyclohexyl
- cyclopentylpurine
- cyclopentylpurine dihydrochloride
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Expired - Fee Related
Links
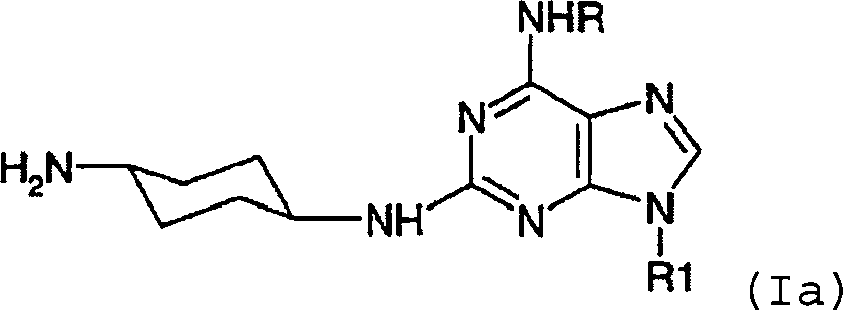
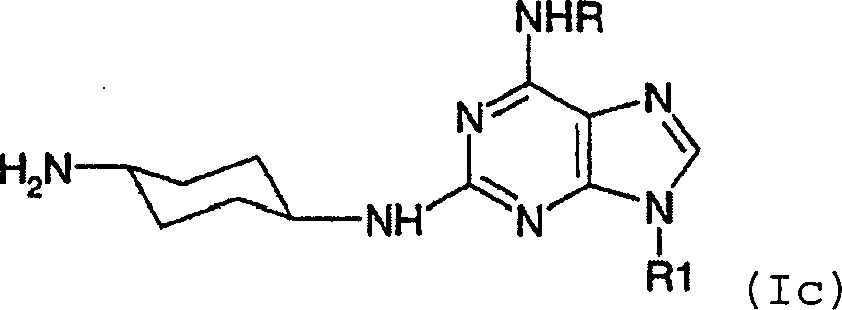
- -1 6,9-DISUBSTITUTED 2- (TRANS- (4-AMINOCYCLOHEXYL) AMINO) PURINE Chemical class 0.000 title claims description 79
- 210000004027 cell Anatomy 0.000 claims description 93
- 150000001875 compounds Chemical class 0.000 claims description 83
- 125000002924 primary amino group Chemical group [H]N([H])* 0.000 claims description 62
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 claims description 46
- 201000010099 disease Diseases 0.000 claims description 38
- 125000001449 isopropyl group Chemical group [H]C([H])([H])C([H])(*)C([H])([H])[H] 0.000 claims description 28
- 150000003839 salts Chemical class 0.000 claims description 25
- 230000006907 apoptotic process Effects 0.000 claims description 23
- 230000003287 optical effect Effects 0.000 claims description 23
- 150000004677 hydrates Chemical class 0.000 claims description 22
- 125000001511 cyclopentyl group Chemical group [H]C1([H])C([H])([H])C([H])([H])C([H])(*)C1([H])[H] 0.000 claims description 21
- 208000032839 leukemia Diseases 0.000 claims description 20
- 125000001424 substituent group Chemical group 0.000 claims description 20
- 201000009030 Carcinoma Diseases 0.000 claims description 19
- 201000001441 melanoma Diseases 0.000 claims description 18
- 238000011282 treatment Methods 0.000 claims description 18
- 230000001613 neoplastic effect Effects 0.000 claims description 16
- UFHFLCQGNIYNRP-UHFFFAOYSA-N Hydrogen Chemical compound [H][H] UFHFLCQGNIYNRP-UHFFFAOYSA-N 0.000 claims description 15
- 239000001257 hydrogen Substances 0.000 claims description 15
- 229910052739 hydrogen Inorganic materials 0.000 claims description 15
- KDCGOANMDULRCW-UHFFFAOYSA-N 7H-purine Chemical compound N1=CNC2=NC=NC2=C1 KDCGOANMDULRCW-UHFFFAOYSA-N 0.000 claims description 14
- 239000002246 antineoplastic agent Substances 0.000 claims description 13
- 210000002569 neuron Anatomy 0.000 claims description 12
- 208000009956 adenocarcinoma Diseases 0.000 claims description 11
- 210000001072 colon Anatomy 0.000 claims description 11
- 239000008194 pharmaceutical composition Substances 0.000 claims description 11
- 239000003814 drug Substances 0.000 claims description 10
- 239000000203 mixture Substances 0.000 claims description 10
- 206010039491 Sarcoma Diseases 0.000 claims description 9
- 125000000217 alkyl group Chemical group 0.000 claims description 9
- 210000000481 breast Anatomy 0.000 claims description 9
- 210000004072 lung Anatomy 0.000 claims description 9
- 125000000753 cycloalkyl group Chemical group 0.000 claims description 8
- 230000006378 damage Effects 0.000 claims description 8
- 239000003937 drug carrier Substances 0.000 claims description 8
- 125000000623 heterocyclic group Chemical group 0.000 claims description 8
- 230000002401 inhibitory effect Effects 0.000 claims description 8
- 210000001672 ovary Anatomy 0.000 claims description 8
- 239000000546 pharmaceutical excipient Substances 0.000 claims description 8
- 125000001997 phenyl group Chemical group [H]C1=C([H])C([H])=C(*)C([H])=C1[H] 0.000 claims description 8
- 125000002947 alkylene group Chemical group 0.000 claims description 7
- 229940034982 antineoplastic agent Drugs 0.000 claims description 7
- 208000035475 disorder Diseases 0.000 claims description 7
- 210000002307 prostate Anatomy 0.000 claims description 7
- 208000017604 Hodgkin disease Diseases 0.000 claims description 6
- 208000010747 Hodgkins lymphoma Diseases 0.000 claims description 6
- 208000026106 cerebrovascular disease Diseases 0.000 claims description 6
- 210000000813 small intestine Anatomy 0.000 claims description 6
- 210000002784 stomach Anatomy 0.000 claims description 6
- 206010024612 Lipoma Diseases 0.000 claims description 5
- 125000000440 benzylamino group Chemical group [H]N(*)C([H])([H])C1=C([H])C([H])=C([H])C([H])=C1[H] 0.000 claims description 5
- 230000003463 hyperproliferative effect Effects 0.000 claims description 5
- 238000004519 manufacturing process Methods 0.000 claims description 5
- 201000008968 osteosarcoma Diseases 0.000 claims description 5
- 208000024893 Acute lymphoblastic leukemia Diseases 0.000 claims description 4
- 208000014697 Acute lymphocytic leukaemia Diseases 0.000 claims description 4
- DTTAQHTWXUWKFN-HEWGHVJBSA-N Cl.Cl.CCCCCCCCNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 Chemical compound Cl.Cl.CCCCCCCCNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 DTTAQHTWXUWKFN-HEWGHVJBSA-N 0.000 claims description 4
- KOQDEGHWASLJDM-VAJGKAQFSA-N Cl.Cl.CCCCCCNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 Chemical compound Cl.Cl.CCCCCCNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 KOQDEGHWASLJDM-VAJGKAQFSA-N 0.000 claims description 4
- HNGBMRQPHQOSAV-HLDGVKIBSA-N Cl.Cl.CCCCCNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 Chemical compound Cl.Cl.CCCCCNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 HNGBMRQPHQOSAV-HLDGVKIBSA-N 0.000 claims description 4
- JIKSKYPICFHQQM-MXPSUWBQSA-N Cl.Cl.CCCCNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C(C)C Chemical compound Cl.Cl.CCCCNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C(C)C JIKSKYPICFHQQM-MXPSUWBQSA-N 0.000 claims description 4
- UFPFCOLXJWMIJS-MXPSUWBQSA-N Cl.Cl.CCCNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 Chemical compound Cl.Cl.CCCNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 UFPFCOLXJWMIJS-MXPSUWBQSA-N 0.000 claims description 4
- HMQFJQYBJAJXTM-MQQSYLBWSA-N Cl.Cl.CCNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 Chemical compound Cl.Cl.CCNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 HMQFJQYBJAJXTM-MQQSYLBWSA-N 0.000 claims description 4
- CPCWYYBYFJMAOX-VAJGKAQFSA-N Cl.Cl.N1=C(N[C@@H]2CC[C@@H](N)CC2)N=C2N(C(C)C)C=NC2=C1NCC1=CC=CC(Cl)=C1 Chemical compound Cl.Cl.N1=C(N[C@@H]2CC[C@@H](N)CC2)N=C2N(C(C)C)C=NC2=C1NCC1=CC=CC(Cl)=C1 CPCWYYBYFJMAOX-VAJGKAQFSA-N 0.000 claims description 4
- CEGZVKSSYGNSFE-MXPSUWBQSA-N Cl.Cl.N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C(C)C)NCC1CC1 Chemical compound Cl.Cl.N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C(C)C)NCC1CC1 CEGZVKSSYGNSFE-MXPSUWBQSA-N 0.000 claims description 4
- YATQQXDPLMIYAK-OPPVHOIFSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCCCCc2ccccc2)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCCCCc2ccccc2)c2ncn(C3CCCC3)c2n1 YATQQXDPLMIYAK-OPPVHOIFSA-N 0.000 claims description 4
- 206010061216 Infarction Diseases 0.000 claims description 4
- 208000006664 Precursor Cell Lymphoblastic Leukemia-Lymphoma Diseases 0.000 claims description 4
- 125000000031 ethylamino group Chemical group [H]C([H])([H])C([H])([H])N([H])[*] 0.000 claims description 4
- 201000011066 hemangioma Diseases 0.000 claims description 4
- 230000007574 infarction Effects 0.000 claims description 4
- 125000005871 1,3-benzodioxolyl group Chemical group 0.000 claims description 3
- 208000031261 Acute myeloid leukaemia Diseases 0.000 claims description 3
- 201000003076 Angiosarcoma Diseases 0.000 claims description 3
- XAZFXDWGSHDJFY-BCHJJPDRSA-N Cl.Cl.CCCCNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 Chemical compound Cl.Cl.CCCCNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 XAZFXDWGSHDJFY-BCHJJPDRSA-N 0.000 claims description 3
- PYRMLGACLIHOFQ-KBTGPXOVSA-N Cl.Cl.CNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 Chemical compound Cl.Cl.CNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 PYRMLGACLIHOFQ-KBTGPXOVSA-N 0.000 claims description 3
- SKMJCTNHPJIHFB-HEWGHVJBSA-N Cl.Cl.Cc1cccc(CNc2nc(N[C@H]3CC[C@H](N)CC3)nc3n(cnc23)C2CCCC2)c1 Chemical compound Cl.Cl.Cc1cccc(CNc2nc(N[C@H]3CC[C@H](N)CC3)nc3n(cnc23)C2CCCC2)c1 SKMJCTNHPJIHFB-HEWGHVJBSA-N 0.000 claims description 3
- OJNFWLMJIGJESU-VAJGKAQFSA-N Cl.Cl.N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)N(CC)C1=CC=NC=C1 Chemical compound Cl.Cl.N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)N(CC)C1=CC=NC=C1 OJNFWLMJIGJESU-VAJGKAQFSA-N 0.000 claims description 3
- GOSNOIRWYFBURZ-BCHJJPDRSA-N Cl.Cl.N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC1CC1 Chemical compound Cl.Cl.N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC1CC1 GOSNOIRWYFBURZ-BCHJJPDRSA-N 0.000 claims description 3
- DJQGGUZDTFVYPH-PMXFUEHMSA-N Cl.Cl.N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC1CCCCC1 Chemical compound Cl.Cl.N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC1CCCCC1 DJQGGUZDTFVYPH-PMXFUEHMSA-N 0.000 claims description 3
- OMADIOWXOWCQMP-ADUSHPDTSA-N Cl.Cl.N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC1OCCCC1 Chemical compound Cl.Cl.N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC1OCCCC1 OMADIOWXOWCQMP-ADUSHPDTSA-N 0.000 claims description 3
- XHLFMOZEGXOWOK-HLDGVKIBSA-N Cl.Cl.N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC=1NC2=C(N1)C=CC=C2 Chemical compound Cl.Cl.N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC=1NC2=C(N1)C=CC=C2 XHLFMOZEGXOWOK-HLDGVKIBSA-N 0.000 claims description 3
- LIVJACUSMIQPGQ-KBTGPXOVSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCC(F)(F)F)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCC(F)(F)F)c2ncn(C3CCCC3)c2n1 LIVJACUSMIQPGQ-KBTGPXOVSA-N 0.000 claims description 3
- CEWOECDKSRZRLK-HLDGVKIBSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCCCCCO)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCCCCCO)c2ncn(C3CCCC3)c2n1 CEWOECDKSRZRLK-HLDGVKIBSA-N 0.000 claims description 3
- XGNNKZFTSOIYTF-MXPSUWBQSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCCCO)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCCCO)c2ncn(C3CCCC3)c2n1 XGNNKZFTSOIYTF-MXPSUWBQSA-N 0.000 claims description 3
- LBQXKDAIVUUZBM-HLDGVKIBSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2ccc(Cl)c(Cl)c2)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2ccc(Cl)c(Cl)c2)c2ncn(C3CCCC3)c2n1 LBQXKDAIVUUZBM-HLDGVKIBSA-N 0.000 claims description 3
- FHMFEMVACWRGCA-VAJGKAQFSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2ccccc2Cl)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2ccccc2Cl)c2ncn(C3CCCC3)c2n1 FHMFEMVACWRGCA-VAJGKAQFSA-N 0.000 claims description 3
- QQRPCIFMRCLLFM-KBTGPXOVSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NNCC(F)(F)F)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NNCC(F)(F)F)c2ncn(C3CCCC3)c2n1 QQRPCIFMRCLLFM-KBTGPXOVSA-N 0.000 claims description 3
- QLARVMUCWBIXSO-MQQSYLBWSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NNCCO)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NNCCO)c2ncn(C3CCCC3)c2n1 QLARVMUCWBIXSO-MQQSYLBWSA-N 0.000 claims description 3
- BNVJNCXUZKAFTF-HEWGHVJBSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NNCCc2ccccc2)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NNCCc2ccccc2)c2ncn(C3CCCC3)c2n1 BNVJNCXUZKAFTF-HEWGHVJBSA-N 0.000 claims description 3
- 208000001258 Hemangiosarcoma Diseases 0.000 claims description 3
- 125000003785 benzimidazolyl group Chemical group N1=C(NC2=C1C=CC=C2)* 0.000 claims description 3
- 210000003679 cervix uteri Anatomy 0.000 claims description 3
- 230000001684 chronic effect Effects 0.000 claims description 3
- 125000000532 dioxanyl group Chemical group 0.000 claims description 3
- 210000003238 esophagus Anatomy 0.000 claims description 3
- 125000002541 furyl group Chemical group 0.000 claims description 3
- 125000002883 imidazolyl group Chemical group 0.000 claims description 3
- 125000001041 indolyl group Chemical group 0.000 claims description 3
- 125000002183 isoquinolinyl group Chemical group C1(=NC=CC2=CC=CC=C12)* 0.000 claims description 3
- 125000000250 methylamino group Chemical group [H]N(*)C([H])([H])[H] 0.000 claims description 3
- 125000002757 morpholinyl group Chemical group 0.000 claims description 3
- 125000002971 oxazolyl group Chemical group 0.000 claims description 3
- 125000004193 piperazinyl group Chemical group 0.000 claims description 3
- 125000003386 piperidinyl group Chemical group 0.000 claims description 3
- 125000000561 purinyl group Chemical group N1=C(N=C2N=CNC2=C1)* 0.000 claims description 3
- 125000004309 pyranyl group Chemical group O1C(C=CC=C1)* 0.000 claims description 3
- 125000003373 pyrazinyl group Chemical group 0.000 claims description 3
- 125000004076 pyridyl group Chemical group 0.000 claims description 3
- 125000000714 pyrimidinyl group Chemical group 0.000 claims description 3
- 125000000719 pyrrolidinyl group Chemical group 0.000 claims description 3
- 125000000168 pyrrolyl group Chemical group 0.000 claims description 3
- 125000002943 quinolinyl group Chemical group N1=C(C=CC2=CC=CC=C12)* 0.000 claims description 3
- 125000003718 tetrahydrofuranyl group Chemical group 0.000 claims description 3
- 125000003507 tetrahydrothiofenyl group Chemical group 0.000 claims description 3
- 125000000335 thiazolyl group Chemical group 0.000 claims description 3
- 125000001544 thienyl group Chemical group 0.000 claims description 3
- 125000004306 triazinyl group Chemical group 0.000 claims description 3
- 101100439046 Caenorhabditis elegans cdk-2 gene Proteins 0.000 claims description 2
- 201000000274 Carcinosarcoma Diseases 0.000 claims description 2
- IRFCBFWZDWZDDZ-HLDGVKIBSA-N Cl.Cl.CC(C)CCNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 Chemical compound Cl.Cl.CC(C)CCNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 IRFCBFWZDWZDDZ-HLDGVKIBSA-N 0.000 claims description 2
- UJHPANQMVMGNNE-RMCIACLISA-N Cl.Cl.CC(CCCC(C)(C)O)Nc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 Chemical compound Cl.Cl.CC(CCCC(C)(C)O)Nc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 UJHPANQMVMGNNE-RMCIACLISA-N 0.000 claims description 2
- FFKQUZRUACEPEH-HEWGHVJBSA-N Cl.Cl.NCCCCCCCCNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 Chemical compound Cl.Cl.NCCCCCCCCNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 FFKQUZRUACEPEH-HEWGHVJBSA-N 0.000 claims description 2
- USLJYPZABXJZCB-BCHJJPDRSA-N Cl.Cl.NCCCCNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 Chemical compound Cl.Cl.NCCCCNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 USLJYPZABXJZCB-BCHJJPDRSA-N 0.000 claims description 2
- 208000033776 Myeloid Acute Leukemia Diseases 0.000 claims description 2
- 125000000842 isoxazolyl group Chemical group 0.000 claims description 2
- 206010024627 liposarcoma Diseases 0.000 claims description 2
- 210000003563 lymphoid tissue Anatomy 0.000 claims description 2
- 230000000684 melanotic effect Effects 0.000 claims description 2
- 125000004573 morpholin-4-yl group Chemical group N1(CCOCC1)* 0.000 claims description 2
- 210000003660 reticulum Anatomy 0.000 claims description 2
- 125000001412 tetrahydropyranyl group Chemical group 0.000 claims description 2
- 125000006308 propyl amino group Chemical group 0.000 claims 2
- JTVILUUAQWQWBK-IYARVYRRSA-N 2-[trans-(4-aminocyclohexyl)amino]-6-(benzyl-amino)-9-cyclopentylpurine Chemical compound C1C[C@@H](N)CC[C@@H]1NC1=NC(NCC=2C=CC=CC=2)=C(N=CN2C3CCCC3)C2=N1 JTVILUUAQWQWBK-IYARVYRRSA-N 0.000 claims 1
- 125000004105 2-pyridyl group Chemical group N1=C([*])C([H])=C([H])C([H])=C1[H] 0.000 claims 1
- 125000003852 3-chlorobenzyl group Chemical group [H]C1=C([H])C(=C([H])C(Cl)=C1[H])C([H])([H])* 0.000 claims 1
- 125000006482 3-iodobenzyl group Chemical group [H]C1=C([H])C(=C([H])C(I)=C1[H])C([H])([H])* 0.000 claims 1
- 125000003349 3-pyridyl group Chemical group N1=C([H])C([*])=C([H])C([H])=C1[H] 0.000 claims 1
- 125000006283 4-chlorobenzyl group Chemical group [H]C1=C([H])C(=C([H])C([H])=C1Cl)C([H])([H])* 0.000 claims 1
- 125000004176 4-fluorobenzyl group Chemical group [H]C1=C([H])C(=C([H])C([H])=C1F)C([H])([H])* 0.000 claims 1
- 125000004203 4-hydroxyphenyl group Chemical group [H]OC1=C([H])C([H])=C(*)C([H])=C1[H] 0.000 claims 1
- 125000000339 4-pyridyl group Chemical group N1=C([H])C([H])=C([*])C([H])=C1[H] 0.000 claims 1
- XDKPTYKTFBDFFM-UHFFFAOYSA-N 9-cyclopentylpurine Chemical compound C1CCCC1N1C2=NC=NC=C2N=C1 XDKPTYKTFBDFFM-UHFFFAOYSA-N 0.000 claims 1
- PXGAFCPCBLPBTK-KHZNEEPTSA-N Cl.Cl.C[C@@H](Nc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1)c1ccccc1 Chemical compound Cl.Cl.C[C@@H](Nc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1)c1ccccc1 PXGAFCPCBLPBTK-KHZNEEPTSA-N 0.000 claims 1
- PXGAFCPCBLPBTK-YLKODNMASA-N Cl.Cl.C[C@H](Nc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1)c1ccccc1 Chemical compound Cl.Cl.C[C@H](Nc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1)c1ccccc1 PXGAFCPCBLPBTK-YLKODNMASA-N 0.000 claims 1
- MTGWFXWSTIXKNA-HLDGVKIBSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCCCc2ncc[nH]2)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCCCc2ncc[nH]2)c2ncn(C3CCCC3)c2n1 MTGWFXWSTIXKNA-HLDGVKIBSA-N 0.000 claims 1
- FMOYTXVEEVGOIG-PMXFUEHMSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2cccc(I)c2)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2cccc(I)c2)c2ncn(C3CCCC3)c2n1 FMOYTXVEEVGOIG-PMXFUEHMSA-N 0.000 claims 1
- 208000006431 amelanotic melanoma Diseases 0.000 claims 1
- 208000025113 myeloid leukemia Diseases 0.000 claims 1
- ZMANZCXQSJIPKH-UHFFFAOYSA-N Triethylamine Chemical compound CCN(CC)CC ZMANZCXQSJIPKH-UHFFFAOYSA-N 0.000 description 174
- 206010028980 Neoplasm Diseases 0.000 description 140
- 238000000262 chemical ionisation mass spectrometry Methods 0.000 description 57
- TVGDBGPLYZULGD-UHFFFAOYSA-N 2,6-dichloro-9-cyclopentylpurine Chemical compound C12=NC(Cl)=NC(Cl)=C2N=CN1C1CCCC1 TVGDBGPLYZULGD-UHFFFAOYSA-N 0.000 description 51
- 108050006400 Cyclin Proteins 0.000 description 33
- 102000016736 Cyclin Human genes 0.000 description 32
- 108090000623 proteins and genes Proteins 0.000 description 32
- 101100005789 Caenorhabditis elegans cdk-4 gene Proteins 0.000 description 28
- 201000011510 cancer Diseases 0.000 description 27
- 238000000034 method Methods 0.000 description 25
- 102100024458 Cyclin-dependent kinase inhibitor 2A Human genes 0.000 description 20
- 235000018102 proteins Nutrition 0.000 description 20
- 102000004169 proteins and genes Human genes 0.000 description 20
- 238000003556 assay Methods 0.000 description 19
- 230000000694 effects Effects 0.000 description 19
- 210000001519 tissue Anatomy 0.000 description 19
- 102000006311 Cyclin D1 Human genes 0.000 description 18
- 108010058546 Cyclin D1 Proteins 0.000 description 17
- 206010006187 Breast cancer Diseases 0.000 description 16
- 101150012716 CDK1 gene Proteins 0.000 description 15
- 101100059559 Emericella nidulans (strain FGSC A4 / ATCC 38163 / CBS 112.46 / NRRL 194 / M139) nimX gene Proteins 0.000 description 15
- 108091000080 Phosphotransferase Proteins 0.000 description 15
- 102000020233 phosphotransferase Human genes 0.000 description 15
- 108020004414 DNA Proteins 0.000 description 14
- 206010025323 Lymphomas Diseases 0.000 description 14
- 206010059394 acanthoma Diseases 0.000 description 14
- 230000035755 proliferation Effects 0.000 description 14
- 208000026310 Breast neoplasm Diseases 0.000 description 13
- 102100036883 Cyclin-H Human genes 0.000 description 13
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 13
- 230000018199 S phase Effects 0.000 description 13
- 230000022131 cell cycle Effects 0.000 description 13
- 229940088597 hormone Drugs 0.000 description 13
- 239000005556 hormone Substances 0.000 description 13
- 210000004881 tumor cell Anatomy 0.000 description 13
- OKKJLVBELUTLKV-UHFFFAOYSA-N Methanol Chemical compound OC OKKJLVBELUTLKV-UHFFFAOYSA-N 0.000 description 12
- 241000700605 Viruses Species 0.000 description 12
- 230000003211 malignant effect Effects 0.000 description 12
- 230000035772 mutation Effects 0.000 description 12
- GTVPOLSIJWJJNY-UHFFFAOYSA-N olomoucine Chemical compound N1=C(NCCO)N=C2N(C)C=NC2=C1NCC1=CC=CC=C1 GTVPOLSIJWJJNY-UHFFFAOYSA-N 0.000 description 12
- 101150073031 cdk2 gene Proteins 0.000 description 11
- 108090000257 Cyclin E Proteins 0.000 description 10
- 102000003909 Cyclin E Human genes 0.000 description 10
- 241000238631 Hexapoda Species 0.000 description 10
- 208000035269 cancer or benign tumor Diseases 0.000 description 10
- 238000011161 development Methods 0.000 description 10
- 230000018109 developmental process Effects 0.000 description 10
- 230000012010 growth Effects 0.000 description 10
- 238000002360 preparation method Methods 0.000 description 10
- 102000002427 Cyclin B Human genes 0.000 description 9
- YMWUJEATGCHHMB-UHFFFAOYSA-N Dichloromethane Chemical compound ClCCl YMWUJEATGCHHMB-UHFFFAOYSA-N 0.000 description 9
- 206010027476 Metastases Diseases 0.000 description 9
- 101100273808 Xenopus laevis cdk1-b gene Proteins 0.000 description 9
- 230000015572 biosynthetic process Effects 0.000 description 9
- 230000026731 phosphorylation Effects 0.000 description 9
- 238000006366 phosphorylation reaction Methods 0.000 description 9
- RMFWVOLULURGJI-UHFFFAOYSA-N 2,6-dichloro-7h-purine Chemical compound ClC1=NC(Cl)=C2NC=NC2=N1 RMFWVOLULURGJI-UHFFFAOYSA-N 0.000 description 8
- GKEFDRUWUYSFGW-UHFFFAOYSA-N 2,6-dichloro-9-propan-2-ylpurine Chemical compound N1=C(Cl)N=C2N(C(C)C)C=NC2=C1Cl GKEFDRUWUYSFGW-UHFFFAOYSA-N 0.000 description 8
- KLWPJMFMVPTNCC-UHFFFAOYSA-N Camptothecin Natural products CCC1(O)C(=O)OCC2=C1C=C3C4Nc5ccccc5C=C4CN3C2=O KLWPJMFMVPTNCC-UHFFFAOYSA-N 0.000 description 8
- 108010068150 Cyclin B Proteins 0.000 description 8
- 108010068237 Cyclin H Proteins 0.000 description 8
- NBIIXXVUZAFLBC-UHFFFAOYSA-N Phosphoric acid Chemical compound OP(O)(O)=O NBIIXXVUZAFLBC-UHFFFAOYSA-N 0.000 description 8
- 229940127093 camptothecin Drugs 0.000 description 8
- VSJKWCGYPAHWDS-FQEVSTJZSA-N camptothecin Chemical compound C1=CC=C2C=C(CN3C4=CC5=C(C3=O)COC(=O)[C@]5(O)CC)C4=NC2=C1 VSJKWCGYPAHWDS-FQEVSTJZSA-N 0.000 description 8
- 230000032823 cell division Effects 0.000 description 8
- UQHKFADEQIVWID-UHFFFAOYSA-N cytokinin Natural products C1=NC=2C(NCC=C(CO)C)=NC=NC=2N1C1CC(O)C(CO)O1 UQHKFADEQIVWID-UHFFFAOYSA-N 0.000 description 8
- 239000004062 cytokinin Substances 0.000 description 8
- VSJKWCGYPAHWDS-UHFFFAOYSA-N dl-camptothecin Natural products C1=CC=C2C=C(CN3C4=CC5=C(C3=O)COC(=O)C5(O)CC)C4=NC2=C1 VSJKWCGYPAHWDS-UHFFFAOYSA-N 0.000 description 8
- 230000014509 gene expression Effects 0.000 description 8
- 210000003734 kidney Anatomy 0.000 description 8
- 210000000056 organ Anatomy 0.000 description 8
- 238000003757 reverse transcription PCR Methods 0.000 description 8
- BTIHMVBBUGXLCJ-OAHLLOKOSA-N seliciclib Chemical compound C=12N=CN(C(C)C)C2=NC(N[C@@H](CO)CC)=NC=1NCC1=CC=CC=C1 BTIHMVBBUGXLCJ-OAHLLOKOSA-N 0.000 description 8
- PUZPDOWCWNUUKD-UHFFFAOYSA-M sodium fluoride Chemical compound [F-].[Na+] PUZPDOWCWNUUKD-UHFFFAOYSA-M 0.000 description 8
- 239000000243 solution Substances 0.000 description 8
- 239000000758 substrate Substances 0.000 description 8
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 7
- 206010035226 Plasma cell myeloma Diseases 0.000 description 7
- 102000001253 Protein Kinase Human genes 0.000 description 7
- 230000002159 abnormal effect Effects 0.000 description 7
- 210000000988 bone and bone Anatomy 0.000 description 7
- 239000002775 capsule Substances 0.000 description 7
- 101150059448 cdk7 gene Proteins 0.000 description 7
- 238000006243 chemical reaction Methods 0.000 description 7
- 208000029742 colonic neoplasm Diseases 0.000 description 7
- 230000004069 differentiation Effects 0.000 description 7
- 230000005764 inhibitory process Effects 0.000 description 7
- 239000002609 medium Substances 0.000 description 7
- 230000000394 mitotic effect Effects 0.000 description 7
- 108090000765 processed proteins & peptides Proteins 0.000 description 7
- 108060006633 protein kinase Proteins 0.000 description 7
- 230000002889 sympathetic effect Effects 0.000 description 7
- 208000011580 syndromic disease Diseases 0.000 description 7
- 238000003786 synthesis reaction Methods 0.000 description 7
- 238000002560 therapeutic procedure Methods 0.000 description 7
- TUIWMOHSPXIADT-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-(1-phenylethyl)purin-6-amine Chemical compound C=1C=CC=CC=1C(C)NC(C=1N=C2)=NC(Cl)=NC=1N2C1CCCC1 TUIWMOHSPXIADT-UHFFFAOYSA-N 0.000 description 6
- QTBSBXVTEAMEQO-UHFFFAOYSA-N Acetic acid Chemical compound CC(O)=O QTBSBXVTEAMEQO-UHFFFAOYSA-N 0.000 description 6
- 102100023995 Beta-nerve growth factor Human genes 0.000 description 6
- 208000011691 Burkitt lymphomas Diseases 0.000 description 6
- 108010060385 Cyclin B1 Proteins 0.000 description 6
- XEKOWRVHYACXOJ-UHFFFAOYSA-N Ethyl acetate Chemical compound CCOC(C)=O XEKOWRVHYACXOJ-UHFFFAOYSA-N 0.000 description 6
- 102100032340 G2/mitotic-specific cyclin-B1 Human genes 0.000 description 6
- 206010058467 Lung neoplasm malignant Diseases 0.000 description 6
- 208000034578 Multiple myelomas Diseases 0.000 description 6
- 208000014767 Myeloproliferative disease Diseases 0.000 description 6
- 108010025020 Nerve Growth Factor Proteins 0.000 description 6
- 208000015914 Non-Hodgkin lymphomas Diseases 0.000 description 6
- 208000002774 Paraproteinemias Diseases 0.000 description 6
- RJKFOVLPORLFTN-LEKSSAKUSA-N Progesterone Chemical compound C1CC2=CC(=O)CC[C@]2(C)[C@@H]2[C@@H]1[C@@H]1CC[C@H](C(=O)C)[C@@]1(C)CC2 RJKFOVLPORLFTN-LEKSSAKUSA-N 0.000 description 6
- NKANXQFJJICGDU-QPLCGJKRSA-N Tamoxifen Chemical compound C=1C=CC=CC=1C(/CC)=C(C=1C=CC(OCCN(C)C)=CC=1)/C1=CC=CC=C1 NKANXQFJJICGDU-QPLCGJKRSA-N 0.000 description 6
- 208000027418 Wounds and injury Diseases 0.000 description 6
- BIIVYFLTOXDAOV-YVEFUNNKSA-N alvocidib Chemical compound O[C@@H]1CN(C)CC[C@@H]1C1=C(O)C=C(O)C2=C1OC(C=1C(=CC=CC=1)Cl)=CC2=O BIIVYFLTOXDAOV-YVEFUNNKSA-N 0.000 description 6
- 229950010817 alvocidib Drugs 0.000 description 6
- 239000000872 buffer Substances 0.000 description 6
- 230000030833 cell death Effects 0.000 description 6
- 230000001419 dependent effect Effects 0.000 description 6
- 208000015181 infectious disease Diseases 0.000 description 6
- 208000014674 injury Diseases 0.000 description 6
- 210000004185 liver Anatomy 0.000 description 6
- 201000005202 lung cancer Diseases 0.000 description 6
- 208000020816 lung neoplasm Diseases 0.000 description 6
- VLKZOEOYAKHREP-UHFFFAOYSA-N n-Hexane Chemical compound CCCCCC VLKZOEOYAKHREP-UHFFFAOYSA-N 0.000 description 6
- 229940053128 nerve growth factor Drugs 0.000 description 6
- 230000001537 neural effect Effects 0.000 description 6
- 230000002062 proliferating effect Effects 0.000 description 6
- 201000005825 prostate adenocarcinoma Diseases 0.000 description 6
- 230000035897 transcription Effects 0.000 description 6
- 238000013518 transcription Methods 0.000 description 6
- JKMHFZQWWAIEOD-UHFFFAOYSA-N 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic acid Chemical compound OCC[NH+]1CCN(CCS([O-])(=O)=O)CC1 JKMHFZQWWAIEOD-UHFFFAOYSA-N 0.000 description 5
- 206010000830 Acute leukaemia Diseases 0.000 description 5
- 206010018338 Glioma Diseases 0.000 description 5
- 241000699670 Mus sp. Species 0.000 description 5
- 230000000118 anti-neoplastic effect Effects 0.000 description 5
- 229940041181 antineoplastic drug Drugs 0.000 description 5
- 210000001185 bone marrow Anatomy 0.000 description 5
- 230000001413 cellular effect Effects 0.000 description 5
- 210000000349 chromosome Anatomy 0.000 description 5
- 229940079593 drug Drugs 0.000 description 5
- 102000015694 estrogen receptors Human genes 0.000 description 5
- 108010038795 estrogen receptors Proteins 0.000 description 5
- 201000004108 hypersplenism Diseases 0.000 description 5
- 230000007246 mechanism Effects 0.000 description 5
- 230000035773 mitosis phase Effects 0.000 description 5
- 206010051747 multiple endocrine neoplasia Diseases 0.000 description 5
- 210000005170 neoplastic cell Anatomy 0.000 description 5
- 230000001105 regulatory effect Effects 0.000 description 5
- 238000012216 screening Methods 0.000 description 5
- 210000002966 serum Anatomy 0.000 description 5
- 239000007787 solid Substances 0.000 description 5
- 239000002904 solvent Substances 0.000 description 5
- 238000001356 surgical procedure Methods 0.000 description 5
- 208000030507 AIDS Diseases 0.000 description 4
- 208000032791 BCR-ABL1 positive chronic myelogenous leukemia Diseases 0.000 description 4
- 206010004146 Basal cell carcinoma Diseases 0.000 description 4
- 108091007914 CDKs Proteins 0.000 description 4
- 208000010833 Chronic myeloid leukaemia Diseases 0.000 description 4
- PXGAFCPCBLPBTK-MRDWXCOYSA-N Cl.Cl.CC(Nc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1)c1ccccc1 Chemical compound Cl.Cl.CC(Nc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1)c1ccccc1 PXGAFCPCBLPBTK-MRDWXCOYSA-N 0.000 description 4
- 206010009944 Colon cancer Diseases 0.000 description 4
- 108010068192 Cyclin A Proteins 0.000 description 4
- 102000003910 Cyclin D Human genes 0.000 description 4
- 108090000259 Cyclin D Proteins 0.000 description 4
- 102100025191 Cyclin-A2 Human genes 0.000 description 4
- 102000013701 Cyclin-Dependent Kinase 4 Human genes 0.000 description 4
- 108010025464 Cyclin-Dependent Kinase 4 Proteins 0.000 description 4
- DHCLVCXQIBBOPH-UHFFFAOYSA-N Glycerol 2-phosphate Chemical compound OCC(CO)OP(O)(O)=O DHCLVCXQIBBOPH-UHFFFAOYSA-N 0.000 description 4
- 239000007995 HEPES buffer Substances 0.000 description 4
- 101000733249 Homo sapiens Tumor suppressor ARF Proteins 0.000 description 4
- VEXZGXHMUGYJMC-UHFFFAOYSA-N Hydrochloric acid Chemical compound Cl VEXZGXHMUGYJMC-UHFFFAOYSA-N 0.000 description 4
- 102000014150 Interferons Human genes 0.000 description 4
- 108010050904 Interferons Proteins 0.000 description 4
- KFZMGEQAYNKOFK-UHFFFAOYSA-N Isopropanol Chemical compound CC(C)O KFZMGEQAYNKOFK-UHFFFAOYSA-N 0.000 description 4
- 208000007766 Kaposi sarcoma Diseases 0.000 description 4
- 201000003793 Myelodysplastic syndrome Diseases 0.000 description 4
- 208000033761 Myelogenous Chronic BCR-ABL Positive Leukemia Diseases 0.000 description 4
- 235000002637 Nicotiana tabacum Nutrition 0.000 description 4
- 206010060862 Prostate cancer Diseases 0.000 description 4
- 102000009572 RNA Polymerase II Human genes 0.000 description 4
- 108010009460 RNA Polymerase II Proteins 0.000 description 4
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 4
- WYURNTSHIVDZCO-UHFFFAOYSA-N Tetrahydrofuran Chemical compound C1CCOC1 WYURNTSHIVDZCO-UHFFFAOYSA-N 0.000 description 4
- 239000007983 Tris buffer Substances 0.000 description 4
- 208000033559 Waldenström macroglobulinemia Diseases 0.000 description 4
- 230000009471 action Effects 0.000 description 4
- 229910000147 aluminium phosphate Inorganic materials 0.000 description 4
- 150000001413 amino acids Chemical group 0.000 description 4
- WGQKYBSKWIADBV-UHFFFAOYSA-N benzylamine Chemical compound NCC1=CC=CC=C1 WGQKYBSKWIADBV-UHFFFAOYSA-N 0.000 description 4
- 201000008274 breast adenocarcinoma Diseases 0.000 description 4
- 125000004432 carbon atom Chemical group C* 0.000 description 4
- 239000003183 carcinogenic agent Substances 0.000 description 4
- 230000015556 catabolic process Effects 0.000 description 4
- 238000001516 cell proliferation assay Methods 0.000 description 4
- 239000000460 chlorine Substances 0.000 description 4
- 230000004186 co-expression Effects 0.000 description 4
- 201000010897 colon adenocarcinoma Diseases 0.000 description 4
- 238000006731 degradation reaction Methods 0.000 description 4
- 230000002068 genetic effect Effects 0.000 description 4
- 210000003128 head Anatomy 0.000 description 4
- 238000000338 in vitro Methods 0.000 description 4
- 238000000021 kinase assay Methods 0.000 description 4
- 210000004379 membrane Anatomy 0.000 description 4
- 239000012528 membrane Substances 0.000 description 4
- 230000031864 metaphase Effects 0.000 description 4
- 230000011278 mitosis Effects 0.000 description 4
- 206010028537 myelofibrosis Diseases 0.000 description 4
- 229910052757 nitrogen Inorganic materials 0.000 description 4
- 208000002154 non-small cell lung carcinoma Diseases 0.000 description 4
- 230000004043 responsiveness Effects 0.000 description 4
- 229920006395 saturated elastomer Polymers 0.000 description 4
- 239000011775 sodium fluoride Substances 0.000 description 4
- 235000013024 sodium fluoride Nutrition 0.000 description 4
- 210000001685 thyroid gland Anatomy 0.000 description 4
- 230000009466 transformation Effects 0.000 description 4
- RIOQSEWOXXDEQQ-UHFFFAOYSA-N triphenylphosphine Chemical compound C1=CC=CC=C1P(C=1C=CC=CC=1)C1=CC=CC=C1 RIOQSEWOXXDEQQ-UHFFFAOYSA-N 0.000 description 4
- LENZDBCJOHFCAS-UHFFFAOYSA-N tris Chemical compound OCC(N)(CO)CO LENZDBCJOHFCAS-UHFFFAOYSA-N 0.000 description 4
- 210000003932 urinary bladder Anatomy 0.000 description 4
- 230000003612 virological effect Effects 0.000 description 4
- AFWRJOYNLMVZQO-GMFATLNBSA-N (1r,2r,4as,8as)-1-[(1e,3e)-5-hydroxy-3-methylpenta-1,3-dienyl]-2,5,5,8a-tetramethyl-3,4,4a,6,7,8-hexahydro-1h-naphthalen-2-ol Chemical compound CC1(C)CCC[C@]2(C)[C@@H](/C=C/C(=C/CO)/C)[C@](C)(O)CC[C@H]21 AFWRJOYNLMVZQO-GMFATLNBSA-N 0.000 description 3
- VZLJOXNZQHTUAP-UHFFFAOYSA-N 1-(2-chloro-9-cyclopentylpurin-6-yl)-2-(2,2,2-trifluoroethyl)hydrazine Chemical compound C1=NC=2C(NNCC(F)(F)F)=NC(Cl)=NC=2N1C1CCCC1 VZLJOXNZQHTUAP-UHFFFAOYSA-N 0.000 description 3
- PARGUTSGGNGALX-UHFFFAOYSA-N 1-(2-chloro-9-cyclopentylpurin-6-yl)-2-(2-phenylethyl)hydrazine Chemical compound C=12N=CN(C3CCCC3)C2=NC(Cl)=NC=1NNCCC1=CC=CC=C1 PARGUTSGGNGALX-UHFFFAOYSA-N 0.000 description 3
- QIQUJURPOLNLKJ-UHFFFAOYSA-N 2-[2-(2-chloro-9-cyclopentylpurin-6-yl)hydrazinyl]ethanol Chemical compound C1=NC=2C(NNCCO)=NC(Cl)=NC=2N1C1CCCC1 QIQUJURPOLNLKJ-UHFFFAOYSA-N 0.000 description 3
- AXSWQXGTVYORIB-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-(2,2,2-trifluoroethyl)purin-6-amine Chemical compound C1=NC=2C(NCC(F)(F)F)=NC(Cl)=NC=2N1C1CCCC1 AXSWQXGTVYORIB-UHFFFAOYSA-N 0.000 description 3
- WFTHZOPVCADNQV-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-(2-morpholin-4-ylethyl)purin-6-amine Chemical compound C=12N=CN(C3CCCC3)C2=NC(Cl)=NC=1NCCN1CCOCC1 WFTHZOPVCADNQV-UHFFFAOYSA-N 0.000 description 3
- QLJKQAGPGFCSJY-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-(2-phenylethyl)purin-6-amine Chemical compound C=12N=CN(C3CCCC3)C2=NC(Cl)=NC=1NCCC1=CC=CC=C1 QLJKQAGPGFCSJY-UHFFFAOYSA-N 0.000 description 3
- ZEPQCIVFHQMRAS-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-(2-pyridin-2-ylethyl)purin-6-amine Chemical compound C=12N=CN(C3CCCC3)C2=NC(Cl)=NC=1NCCC1=CC=CC=N1 ZEPQCIVFHQMRAS-UHFFFAOYSA-N 0.000 description 3
- JWCYBVYVCDQOQX-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-(3-imidazol-1-ylpropyl)purin-6-amine Chemical compound C=12N=CN(C3CCCC3)C2=NC(Cl)=NC=1NCCCN1C=CN=C1 JWCYBVYVCDQOQX-UHFFFAOYSA-N 0.000 description 3
- ZKDCPTXGSHFZAR-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-(3-methylbutyl)purin-6-amine Chemical compound C1=NC=2C(NCCC(C)C)=NC(Cl)=NC=2N1C1CCCC1 ZKDCPTXGSHFZAR-UHFFFAOYSA-N 0.000 description 3
- NVWIIPRZHFHBCP-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-(3-morpholin-4-ylpropyl)purin-6-amine Chemical compound C=12N=CN(C3CCCC3)C2=NC(Cl)=NC=1NCCCN1CCOCC1 NVWIIPRZHFHBCP-UHFFFAOYSA-N 0.000 description 3
- JRSJDDFYXPJLJF-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-(3-phenylpropyl)purin-6-amine Chemical compound C=12N=CN(C3CCCC3)C2=NC(Cl)=NC=1NCCCC1=CC=CC=C1 JRSJDDFYXPJLJF-UHFFFAOYSA-N 0.000 description 3
- DMFKUKKGUQYKLL-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-(4-phenylbutyl)purin-6-amine Chemical compound C=12N=CN(C3CCCC3)C2=NC(Cl)=NC=1NCCCCC1=CC=CC=C1 DMFKUKKGUQYKLL-UHFFFAOYSA-N 0.000 description 3
- WVXLUVLVNDAKOP-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-(cyclopropylmethyl)purin-6-amine Chemical compound C=12N=CN(C3CCCC3)C2=NC(Cl)=NC=1NCC1CC1 WVXLUVLVNDAKOP-UHFFFAOYSA-N 0.000 description 3
- WYUHRYCZWPJDJD-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-(oxan-2-ylmethyl)purin-6-amine Chemical compound C=12N=CN(C3CCCC3)C2=NC(Cl)=NC=1NCC1CCCCO1 WYUHRYCZWPJDJD-UHFFFAOYSA-N 0.000 description 3
- CSWVTRMSVDQTGK-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-(pyridin-4-ylmethyl)purin-6-amine Chemical compound C=12N=CN(C3CCCC3)C2=NC(Cl)=NC=1NCC1=CC=NC=C1 CSWVTRMSVDQTGK-UHFFFAOYSA-N 0.000 description 3
- KLDAITOUPKPABC-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-[(2,4-dichlorophenyl)methyl]purin-6-amine Chemical compound ClC1=CC(Cl)=CC=C1CNC1=NC(Cl)=NC2=C1N=CN2C1CCCC1 KLDAITOUPKPABC-UHFFFAOYSA-N 0.000 description 3
- KXKRLJZRUPYKSK-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-[(2-fluorophenyl)methyl]purin-6-amine Chemical compound FC1=CC=CC=C1CNC1=NC(Cl)=NC2=C1N=CN2C1CCCC1 KXKRLJZRUPYKSK-UHFFFAOYSA-N 0.000 description 3
- UEYMGJMZPGAFCQ-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-[(3,4-dichlorophenyl)methyl]purin-6-amine Chemical compound C=12N=CN(C3CCCC3)C2=NC(Cl)=NC=1NCC1=CC=C(Cl)C(Cl)=C1 UEYMGJMZPGAFCQ-UHFFFAOYSA-N 0.000 description 3
- VHEUKRCRWNSWES-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-[(3-methylphenyl)methyl]purin-6-amine Chemical compound CC1=CC=CC(CNC=2C=3N=CN(C=3N=C(Cl)N=2)C2CCCC2)=C1 VHEUKRCRWNSWES-UHFFFAOYSA-N 0.000 description 3
- WCFILJOTLUSMLX-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-[(4-fluorophenyl)methyl]purin-6-amine Chemical compound C1=CC(F)=CC=C1CNC1=NC(Cl)=NC2=C1N=CN2C1CCCC1 WCFILJOTLUSMLX-UHFFFAOYSA-N 0.000 description 3
- CEMMPSJINJRHLG-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-ethyl-n-(1h-indol-3-yl)purin-6-amine Chemical compound C=1NC2=CC=CC=C2C=1N(CC)C(C=1N=C2)=NC(Cl)=NC=1N2C1CCCC1 CEMMPSJINJRHLG-UHFFFAOYSA-N 0.000 description 3
- ATCDYOZUIDZNSG-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-ethyl-n-pyridin-3-ylpurin-6-amine Chemical compound N=1C(Cl)=NC=2N(C3CCCC3)C=NC=2C=1N(CC)C1=CC=CN=C1 ATCDYOZUIDZNSG-UHFFFAOYSA-N 0.000 description 3
- XIKHSDSETLHGSH-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-ethyl-n-pyridin-4-ylpurin-6-amine Chemical compound N=1C(Cl)=NC=2N(C3CCCC3)C=NC=2C=1N(CC)C1=CC=NC=C1 XIKHSDSETLHGSH-UHFFFAOYSA-N 0.000 description 3
- OIUGUEQFWXNSPR-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-ethylpurin-6-amine Chemical compound C1=NC=2C(NCC)=NC(Cl)=NC=2N1C1CCCC1 OIUGUEQFWXNSPR-UHFFFAOYSA-N 0.000 description 3
- LHZHRAFIWBWVHJ-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-hexylpurin-6-amine Chemical compound C1=NC=2C(NCCCCCC)=NC(Cl)=NC=2N1C1CCCC1 LHZHRAFIWBWVHJ-UHFFFAOYSA-N 0.000 description 3
- IKYTXKJDCDIVTJ-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-methylpurin-6-amine Chemical compound C1=NC=2C(NC)=NC(Cl)=NC=2N1C1CCCC1 IKYTXKJDCDIVTJ-UHFFFAOYSA-N 0.000 description 3
- VIVHSNXBTRADQA-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-octylpurin-6-amine Chemical compound C1=NC=2C(NCCCCCCCC)=NC(Cl)=NC=2N1C1CCCC1 VIVHSNXBTRADQA-UHFFFAOYSA-N 0.000 description 3
- PWPRDQIWZVWOQX-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-pentylpurin-6-amine Chemical compound C1=NC=2C(NCCCCC)=NC(Cl)=NC=2N1C1CCCC1 PWPRDQIWZVWOQX-UHFFFAOYSA-N 0.000 description 3
- AAVVCMCCIQNHJC-UHFFFAOYSA-N 2-chloro-9-cyclopentyl-n-propylpurin-6-amine Chemical compound C1=NC=2C(NCCC)=NC(Cl)=NC=2N1C1CCCC1 AAVVCMCCIQNHJC-UHFFFAOYSA-N 0.000 description 3
- HRXPDFBFTYMTIC-UHFFFAOYSA-N 2-chloro-n-(cyclohexylmethyl)-9-cyclopentylpurin-6-amine Chemical compound C=12N=CN(C3CCCC3)C2=NC(Cl)=NC=1NCC1CCCCC1 HRXPDFBFTYMTIC-UHFFFAOYSA-N 0.000 description 3
- LUFVCWKGDHIRFB-UHFFFAOYSA-N 2-chloro-n-(cyclopropylmethyl)-9-propan-2-ylpurin-6-amine Chemical compound N1=C(Cl)N=C2N(C(C)C)C=NC2=C1NCC1CC1 LUFVCWKGDHIRFB-UHFFFAOYSA-N 0.000 description 3
- PXOAWFNKQIMZNO-UHFFFAOYSA-N 2-chloro-n-[(2-chlorophenyl)methyl]-9-cyclopentylpurin-6-amine Chemical compound C=12N=CN(C3CCCC3)C2=NC(Cl)=NC=1NCC1=CC=CC=C1Cl PXOAWFNKQIMZNO-UHFFFAOYSA-N 0.000 description 3
- ICVOWBDATODGBX-UHFFFAOYSA-N 2-chloro-n-[(3-chlorophenyl)methyl]-9-cyclopentylpurin-6-amine Chemical compound ClC1=CC=CC(CNC=2C=3N=CN(C=3N=C(Cl)N=2)C2CCCC2)=C1 ICVOWBDATODGBX-UHFFFAOYSA-N 0.000 description 3
- FDNMBIXJMACSQH-UHFFFAOYSA-N 2-chloro-n-[(3-chlorophenyl)methyl]-9-propan-2-ylpurin-6-amine Chemical compound N1=C(Cl)N=C2N(C(C)C)C=NC2=C1NCC1=CC=CC(Cl)=C1 FDNMBIXJMACSQH-UHFFFAOYSA-N 0.000 description 3
- BYCSUOWXFFRLEY-UHFFFAOYSA-N 2-chloro-n-[(3-iodophenyl)methyl]-9-propan-2-ylpurin-6-amine Chemical compound N1=C(Cl)N=C2N(C(C)C)C=NC2=C1NCC1=CC=CC(I)=C1 BYCSUOWXFFRLEY-UHFFFAOYSA-N 0.000 description 3
- SRFPGGQQOCFAMR-UHFFFAOYSA-N 2-chloro-n-[(4-chloro-2-fluorophenyl)methyl]-9-cyclopentylpurin-6-amine Chemical compound FC1=CC(Cl)=CC=C1CNC1=NC(Cl)=NC2=C1N=CN2C1CCCC1 SRFPGGQQOCFAMR-UHFFFAOYSA-N 0.000 description 3
- UDVDHBMJOJPDIW-UHFFFAOYSA-N 2-chloro-n-[(4-chlorophenyl)methyl]-9-cyclopentylpurin-6-amine Chemical compound C1=CC(Cl)=CC=C1CNC1=NC(Cl)=NC2=C1N=CN2C1CCCC1 UDVDHBMJOJPDIW-UHFFFAOYSA-N 0.000 description 3
- GHPMPLIORQAVBY-UHFFFAOYSA-N 3-[(2-chloro-9-cyclopentylpurin-6-yl)amino]propan-1-ol Chemical compound C1=NC=2C(NCCCO)=NC(Cl)=NC=2N1C1CCCC1 GHPMPLIORQAVBY-UHFFFAOYSA-N 0.000 description 3
- CLVMUXVFUCCAMI-UHFFFAOYSA-N 4-[2-[(2-chloro-9-cyclopentylpurin-6-yl)amino]ethyl]phenol Chemical compound C1=CC(O)=CC=C1CCNC1=NC(Cl)=NC2=C1N=CN2C1CCCC1 CLVMUXVFUCCAMI-UHFFFAOYSA-N 0.000 description 3
- AFXLNZZDUGOKEO-UHFFFAOYSA-N 5-[(2-chloro-9-cyclopentylpurin-6-yl)amino]pentan-1-ol Chemical compound C1=NC=2C(NCCCCCO)=NC(Cl)=NC=2N1C1CCCC1 AFXLNZZDUGOKEO-UHFFFAOYSA-N 0.000 description 3
- ITJDNUOTQDBTGF-UHFFFAOYSA-N 6-[(2-chloro-9-cyclopentylpurin-6-yl)amino]-2-methylheptan-2-ol Chemical compound C1=NC=2C(NC(CCCC(C)(C)O)C)=NC(Cl)=NC=2N1C1CCCC1 ITJDNUOTQDBTGF-UHFFFAOYSA-N 0.000 description 3
- WEVYAHXRMPXWCK-UHFFFAOYSA-N Acetonitrile Chemical compound CC#N WEVYAHXRMPXWCK-UHFFFAOYSA-N 0.000 description 3
- 108010085238 Actins Proteins 0.000 description 3
- 102000007469 Actins Human genes 0.000 description 3
- 101150072950 BRCA1 gene Proteins 0.000 description 3
- 206010060999 Benign neoplasm Diseases 0.000 description 3
- UHOVQNZJYSORNB-UHFFFAOYSA-N Benzene Chemical compound C1=CC=CC=C1 UHOVQNZJYSORNB-UHFFFAOYSA-N 0.000 description 3
- 108091003079 Bovine Serum Albumin Proteins 0.000 description 3
- 239000004215 Carbon black (E152) Substances 0.000 description 3
- 102000005483 Cell Cycle Proteins Human genes 0.000 description 3
- 108010031896 Cell Cycle Proteins Proteins 0.000 description 3
- 208000005243 Chondrosarcoma Diseases 0.000 description 3
- ALHUQAMESCFYKP-VAJGKAQFSA-N Cl.Cl.CC(C)n1cnc2c(NCc3cccc(I)c3)nc(N[C@H]3CC[C@H](N)CC3)nc12 Chemical compound Cl.Cl.CC(C)n1cnc2c(NCc3cccc(I)c3)nc(N[C@H]3CC[C@H](N)CC3)nc12 ALHUQAMESCFYKP-VAJGKAQFSA-N 0.000 description 3
- 102000009512 Cyclin-Dependent Kinase Inhibitor p15 Human genes 0.000 description 3
- 108010009356 Cyclin-Dependent Kinase Inhibitor p15 Proteins 0.000 description 3
- 102000003903 Cyclin-dependent kinases Human genes 0.000 description 3
- 108090000266 Cyclin-dependent kinases Proteins 0.000 description 3
- 230000006820 DNA synthesis Effects 0.000 description 3
- 206010014759 Endometrial neoplasm Diseases 0.000 description 3
- LYCAIKOWRPUZTN-UHFFFAOYSA-N Ethylene glycol Chemical compound OCCO LYCAIKOWRPUZTN-UHFFFAOYSA-N 0.000 description 3
- 201000008808 Fibrosarcoma Diseases 0.000 description 3
- 229940123414 Folate antagonist Drugs 0.000 description 3
- 208000032612 Glial tumor Diseases 0.000 description 3
- PEDCQBHIVMGVHV-UHFFFAOYSA-N Glycerine Chemical compound OCC(O)CO PEDCQBHIVMGVHV-UHFFFAOYSA-N 0.000 description 3
- 101000980932 Homo sapiens Cyclin-dependent kinase inhibitor 2A Proteins 0.000 description 3
- 241000701044 Human gammaherpesvirus 4 Species 0.000 description 3
- 102000006835 Lamins Human genes 0.000 description 3
- 108010047294 Lamins Proteins 0.000 description 3
- 206010073099 Lobular breast carcinoma in situ Diseases 0.000 description 3
- 241001465754 Metazoa Species 0.000 description 3
- 108060008487 Myosin Proteins 0.000 description 3
- 102000003505 Myosin Human genes 0.000 description 3
- JGFZNNIVVJXRND-UHFFFAOYSA-N N,N-Diisopropylethylamine (DIPEA) Chemical compound CCN(C(C)C)C(C)C JGFZNNIVVJXRND-UHFFFAOYSA-N 0.000 description 3
- 241000208125 Nicotiana Species 0.000 description 3
- 206010061535 Ovarian neoplasm Diseases 0.000 description 3
- 208000037062 Polyps Diseases 0.000 description 3
- 208000000236 Prostatic Neoplasms Diseases 0.000 description 3
- 210000001744 T-lymphocyte Anatomy 0.000 description 3
- 102000040945 Transcription factor Human genes 0.000 description 3
- 108091023040 Transcription factor Proteins 0.000 description 3
- 229940024606 amino acid Drugs 0.000 description 3
- 235000001014 amino acid Nutrition 0.000 description 3
- 239000011324 bead Substances 0.000 description 3
- 210000004369 blood Anatomy 0.000 description 3
- 239000008280 blood Substances 0.000 description 3
- HQABUPZFAYXKJW-UHFFFAOYSA-N butan-1-amine Chemical compound CCCCN HQABUPZFAYXKJW-UHFFFAOYSA-N 0.000 description 3
- 210000004899 c-terminal region Anatomy 0.000 description 3
- 229910052799 carbon Inorganic materials 0.000 description 3
- 231100000357 carcinogen Toxicity 0.000 description 3
- 239000013592 cell lysate Substances 0.000 description 3
- 238000005119 centrifugation Methods 0.000 description 3
- 230000002490 cerebral effect Effects 0.000 description 3
- 230000008859 change Effects 0.000 description 3
- 239000002299 complementary DNA Substances 0.000 description 3
- 229940043378 cyclin-dependent kinase inhibitor Drugs 0.000 description 3
- 108010072268 cyclin-dependent kinase-activating kinase Proteins 0.000 description 3
- VKIRRGRTJUUZHS-UHFFFAOYSA-N cyclohexane-1,4-diamine Chemical compound NC1CCC(N)CC1 VKIRRGRTJUUZHS-UHFFFAOYSA-N 0.000 description 3
- 230000034994 death Effects 0.000 description 3
- 238000009826 distribution Methods 0.000 description 3
- 230000002124 endocrine Effects 0.000 description 3
- 238000005516 engineering process Methods 0.000 description 3
- 229940011871 estrogen Drugs 0.000 description 3
- 239000000262 estrogen Substances 0.000 description 3
- DEFVIWRASFVYLL-UHFFFAOYSA-N ethylene glycol bis(2-aminoethyl)tetraacetic acid Chemical compound OC(=O)CN(CC(O)=O)CCOCCOCCN(CC(O)=O)CC(O)=O DEFVIWRASFVYLL-UHFFFAOYSA-N 0.000 description 3
- 238000000605 extraction Methods 0.000 description 3
- 210000001035 gastrointestinal tract Anatomy 0.000 description 3
- 210000004602 germ cell Anatomy 0.000 description 3
- 208000035474 group of disease Diseases 0.000 description 3
- 229930195733 hydrocarbon Natural products 0.000 description 3
- 230000002779 inactivation Effects 0.000 description 3
- 239000003112 inhibitor Substances 0.000 description 3
- 210000005053 lamin Anatomy 0.000 description 3
- 201000010260 leiomyoma Diseases 0.000 description 3
- 210000001165 lymph node Anatomy 0.000 description 3
- 201000000564 macroglobulinemia Diseases 0.000 description 3
- 230000001404 mediated effect Effects 0.000 description 3
- 210000002418 meninge Anatomy 0.000 description 3
- 230000009401 metastasis Effects 0.000 description 3
- 210000000865 mononuclear phagocyte system Anatomy 0.000 description 3
- 201000005962 mycosis fungoides Diseases 0.000 description 3
- RXYSYSWNWNBFEW-UHFFFAOYSA-N n'-(2-chloro-9-cyclopentylpurin-6-yl)butane-1,4-diamine Chemical compound C1=NC=2C(NCCCCN)=NC(Cl)=NC=2N1C1CCCC1 RXYSYSWNWNBFEW-UHFFFAOYSA-N 0.000 description 3
- XZJIJUJRPVKWNQ-UHFFFAOYSA-N n'-(2-chloro-9-cyclopentylpurin-6-yl)octane-1,8-diamine Chemical compound C1=NC=2C(NCCCCCCCCN)=NC(Cl)=NC=2N1C1CCCC1 XZJIJUJRPVKWNQ-UHFFFAOYSA-N 0.000 description 3
- TVOBOOZMQVVYQY-UHFFFAOYSA-N n-(1h-benzimidazol-2-ylmethyl)-2-chloro-9-cyclopentylpurin-6-amine Chemical compound C12=NC(Cl)=NC(NCC=3NC4=CC=CC=C4N=3)=C2N=CN1C1CCCC1 TVOBOOZMQVVYQY-UHFFFAOYSA-N 0.000 description 3
- RXKXDCFCCYMBIA-UHFFFAOYSA-N n-[2-(1,3-benzodioxol-5-yl)ethyl]-2-chloro-9-cyclopentylpurin-6-amine Chemical compound C12=NC(Cl)=NC(NCCC=3C=C4OCOC4=CC=3)=C2N=CN1C1CCCC1 RXKXDCFCCYMBIA-UHFFFAOYSA-N 0.000 description 3
- APURIMWVMQIOPD-UHFFFAOYSA-N n-benzyl-2-chloro-9-cyclopentylpurin-6-amine Chemical compound C=12N=CN(C3CCCC3)C2=NC(Cl)=NC=1NCC1=CC=CC=C1 APURIMWVMQIOPD-UHFFFAOYSA-N 0.000 description 3
- NSQQTIIWZCVMRW-UHFFFAOYSA-N n-benzyl-2-chloro-9-propan-2-ylpurin-6-amine Chemical compound N1=C(Cl)N=C2N(C(C)C)C=NC2=C1NCC1=CC=CC=C1 NSQQTIIWZCVMRW-UHFFFAOYSA-N 0.000 description 3
- RELUKSSWENGPFT-UHFFFAOYSA-N n-butyl-2-chloro-9-cyclopentylpurin-6-amine Chemical compound C1=NC=2C(NCCCC)=NC(Cl)=NC=2N1C1CCCC1 RELUKSSWENGPFT-UHFFFAOYSA-N 0.000 description 3
- PVNNWJKFPLRMTP-UHFFFAOYSA-N n-butyl-2-chloro-9-propan-2-ylpurin-6-amine Chemical compound CCCCNC1=NC(Cl)=NC2=C1N=CN2C(C)C PVNNWJKFPLRMTP-UHFFFAOYSA-N 0.000 description 3
- 210000004498 neuroglial cell Anatomy 0.000 description 3
- 238000011580 nude mouse model Methods 0.000 description 3
- 230000002018 overexpression Effects 0.000 description 3
- 230000037361 pathway Effects 0.000 description 3
- 208000028591 pheochromocytoma Diseases 0.000 description 3
- 229940080469 phosphocellulose Drugs 0.000 description 3
- 230000001817 pituitary effect Effects 0.000 description 3
- 208000037244 polycythemia vera Diseases 0.000 description 3
- 208000003476 primary myelofibrosis Diseases 0.000 description 3
- 239000000186 progesterone Substances 0.000 description 3
- 229960003387 progesterone Drugs 0.000 description 3
- 125000001436 propyl group Chemical group [H]C([*])([H])C([H])([H])C([H])([H])[H] 0.000 description 3
- 230000005855 radiation Effects 0.000 description 3
- 239000000376 reactant Substances 0.000 description 3
- 210000000664 rectum Anatomy 0.000 description 3
- 230000022983 regulation of cell cycle Effects 0.000 description 3
- 238000011160 research Methods 0.000 description 3
- 230000000284 resting effect Effects 0.000 description 3
- 210000000278 spinal cord Anatomy 0.000 description 3
- 230000000638 stimulation Effects 0.000 description 3
- 229960001603 tamoxifen Drugs 0.000 description 3
- 230000007704 transition Effects 0.000 description 3
- DTQVDTLACAAQTR-UHFFFAOYSA-N trifluoroacetic acid Substances OC(=O)C(F)(F)F DTQVDTLACAAQTR-UHFFFAOYSA-N 0.000 description 3
- IHIXIJGXTJIKRB-UHFFFAOYSA-N trisodium vanadate Chemical compound [Na+].[Na+].[Na+].[O-][V]([O-])([O-])=O IHIXIJGXTJIKRB-UHFFFAOYSA-N 0.000 description 3
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 3
- BJFPYGGTDAYECS-UHFFFAOYSA-N (3-chlorophenyl)methanamine Chemical compound NCC1=CC=CC(Cl)=C1 BJFPYGGTDAYECS-UHFFFAOYSA-N 0.000 description 2
- 0 *c1c2nc[n](*)c2nc(NC(CC2)CCC2N)n1 Chemical compound *c1c2nc[n](*)c2nc(NC(CC2)CCC2N)n1 0.000 description 2
- 125000004973 1-butenyl group Chemical group C(=CCC)* 0.000 description 2
- 125000006017 1-propenyl group Chemical group 0.000 description 2
- 238000005160 1H NMR spectroscopy Methods 0.000 description 2
- 125000004974 2-butenyl group Chemical group C(C=CC)* 0.000 description 2
- 125000003903 2-propenyl group Chemical group [H]C([*])([H])C([H])=C([H])[H] 0.000 description 2
- 125000004975 3-butenyl group Chemical group C(CC=C)* 0.000 description 2
- XTXXRLKBCNTEFW-UHFFFAOYSA-N 4-[(2-chloro-9-propan-2-ylpurin-6-yl)amino]-1-(1h-imidazol-2-yl)butan-1-one Chemical compound N1=C(Cl)N=C2N(C(C)C)C=NC2=C1NCCCC(=O)C1=NC=CN1 XTXXRLKBCNTEFW-UHFFFAOYSA-N 0.000 description 2
- 108091093088 Amplicon Proteins 0.000 description 2
- PAYRUJLWNCNPSJ-UHFFFAOYSA-N Aniline Chemical compound NC1=CC=CC=C1 PAYRUJLWNCNPSJ-UHFFFAOYSA-N 0.000 description 2
- 102000015790 Asparaginase Human genes 0.000 description 2
- 108010024976 Asparaginase Proteins 0.000 description 2
- IJGRMHOSHXDMSA-UHFFFAOYSA-N Atomic nitrogen Chemical compound N#N IJGRMHOSHXDMSA-UHFFFAOYSA-N 0.000 description 2
- 208000010839 B-cell chronic lymphocytic leukemia Diseases 0.000 description 2
- 108700040618 BRCA1 Genes Proteins 0.000 description 2
- 101100002068 Bacillus subtilis (strain 168) araR gene Proteins 0.000 description 2
- 206010005003 Bladder cancer Diseases 0.000 description 2
- 206010007270 Carcinoid syndrome Diseases 0.000 description 2
- 208000000419 Chronic Hepatitis B Diseases 0.000 description 2
- PGLACAYZXSFZON-VAJGKAQFSA-N Cl.Cl.CC(C)n1cnc2c(NCc3ccccc3)nc(N[C@H]3CC[C@H](N)CC3)nc12 Chemical compound Cl.Cl.CC(C)n1cnc2c(NCc3ccccc3)nc(N[C@H]3CC[C@H](N)CC3)nc12 PGLACAYZXSFZON-VAJGKAQFSA-N 0.000 description 2
- HVWAEQMIESKPPF-SRWQXTDJSA-N Cl.Cl.Cl.N1=C(N[C@@H]2CC[C@@H](N)CC2)N=C2N(C(C)C)C=NC2=C1NCCCC(=O)C1=NC=CN1 Chemical compound Cl.Cl.Cl.N1=C(N[C@@H]2CC[C@@H](N)CC2)N=C2N(C(C)C)C=NC2=C1NCCCC(=O)C1=NC=CN1 HVWAEQMIESKPPF-SRWQXTDJSA-N 0.000 description 2
- FKIRKHQOZSEIOO-XAASAEJFSA-N Cl.Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCCN2CCOCC2)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCCN2CCOCC2)c2ncn(C3CCCC3)c2n1 FKIRKHQOZSEIOO-XAASAEJFSA-N 0.000 description 2
- NDFRQWKMCFIWRF-PMXFUEHMSA-N Cl.Cl.N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)N(CC)C1=CNC2=CC=CC=C12 Chemical compound Cl.Cl.N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)N(CC)C1=CNC2=CC=CC=C12 NDFRQWKMCFIWRF-PMXFUEHMSA-N 0.000 description 2
- VNQVFYGXPJYYQK-VAJGKAQFSA-N Cl.Cl.N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)N(CC)C=1C=NC=CC1 Chemical compound Cl.Cl.N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)N(CC)C=1C=NC=CC1 VNQVFYGXPJYYQK-VAJGKAQFSA-N 0.000 description 2
- NKRGUTZYHYWUSM-VAJGKAQFSA-N Cl.Cl.N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC1=CC=NC=C1 Chemical compound Cl.Cl.N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC1=CC=NC=C1 NKRGUTZYHYWUSM-VAJGKAQFSA-N 0.000 description 2
- KEACBXUHPFOAFG-PMXFUEHMSA-N Cl.Cl.N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCCC1=CC=C(C=C1)O Chemical compound Cl.Cl.N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCCC1=CC=C(C=C1)O KEACBXUHPFOAFG-PMXFUEHMSA-N 0.000 description 2
- ABNYSVHNVWQNLF-PMXFUEHMSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCCCN2CCOCC2)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCCCN2CCOCC2)c2ncn(C3CCCC3)c2n1 ABNYSVHNVWQNLF-PMXFUEHMSA-N 0.000 description 2
- HSPRTNOJKSBQIH-RKQOQJRBSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCCCc2ccccc2)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCCCc2ccccc2)c2ncn(C3CCCC3)c2n1 HSPRTNOJKSBQIH-RKQOQJRBSA-N 0.000 description 2
- AYAGVBGIKQOZOP-VAJGKAQFSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCCCn2ccnc2)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCCCn2ccnc2)c2ncn(C3CCCC3)c2n1 AYAGVBGIKQOZOP-VAJGKAQFSA-N 0.000 description 2
- SQPPFFDJUJNGFI-PMXFUEHMSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCCc2ccc3OCOc3c2)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCCc2ccc3OCOc3c2)c2ncn(C3CCCC3)c2n1 SQPPFFDJUJNGFI-PMXFUEHMSA-N 0.000 description 2
- JXRRLJGBLBSLRJ-HEWGHVJBSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCCc2ccccc2)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCCc2ccccc2)c2ncn(C3CCCC3)c2n1 JXRRLJGBLBSLRJ-HEWGHVJBSA-N 0.000 description 2
- RULVROGKFXYWKJ-PMXFUEHMSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2ccc(Cl)cc2)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2ccc(Cl)cc2)c2ncn(C3CCCC3)c2n1 RULVROGKFXYWKJ-PMXFUEHMSA-N 0.000 description 2
- ZJGSLTSUKHINIW-VAJGKAQFSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2ccc(Cl)cc2Cl)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2ccc(Cl)cc2Cl)c2ncn(C3CCCC3)c2n1 ZJGSLTSUKHINIW-VAJGKAQFSA-N 0.000 description 2
- ZYRNWUOVMMFIBD-VAJGKAQFSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2ccc(Cl)cc2F)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2ccc(Cl)cc2F)c2ncn(C3CCCC3)c2n1 ZYRNWUOVMMFIBD-VAJGKAQFSA-N 0.000 description 2
- ZYGKEJNDNZARNY-PMXFUEHMSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2ccc(F)cc2)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2ccc(F)cc2)c2ncn(C3CCCC3)c2n1 ZYGKEJNDNZARNY-PMXFUEHMSA-N 0.000 description 2
- ZQSCTILOQZDDTI-PMXFUEHMSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2cccc(Cl)c2)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2cccc(Cl)c2)c2ncn(C3CCCC3)c2n1 ZQSCTILOQZDDTI-PMXFUEHMSA-N 0.000 description 2
- LOZMADXGBQKSAL-PMXFUEHMSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2ccccc2)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2ccccc2)c2ncn(C3CCCC3)c2n1 LOZMADXGBQKSAL-PMXFUEHMSA-N 0.000 description 2
- 108020004635 Complementary DNA Proteins 0.000 description 2
- 208000011231 Crohn disease Diseases 0.000 description 2
- 102100033233 Cyclin-dependent kinase inhibitor 1B Human genes 0.000 description 2
- 230000004543 DNA replication Effects 0.000 description 2
- 206010012735 Diarrhoea Diseases 0.000 description 2
- AOJJSUZBOXZQNB-TZSSRYMLSA-N Doxorubicin Chemical compound O([C@H]1C[C@@](O)(CC=2C(O)=C3C(=O)C=4C=CC=C(C=4C(=O)C3=C(O)C=21)OC)C(=O)CO)[C@H]1C[C@H](N)[C@H](O)[C@H](C)O1 AOJJSUZBOXZQNB-TZSSRYMLSA-N 0.000 description 2
- 102000004190 Enzymes Human genes 0.000 description 2
- 108090000790 Enzymes Proteins 0.000 description 2
- 208000006168 Ewing Sarcoma Diseases 0.000 description 2
- 208000007300 Fibrolamellar hepatocellular carcinoma Diseases 0.000 description 2
- 206010016654 Fibrosis Diseases 0.000 description 2
- CEAZRRDELHUEMR-URQXQFDESA-N Gentamicin Chemical compound O1[C@H](C(C)NC)CC[C@@H](N)[C@H]1O[C@H]1[C@H](O)[C@@H](O[C@@H]2[C@@H]([C@@H](NC)[C@@](C)(O)CO2)O)[C@H](N)C[C@@H]1N CEAZRRDELHUEMR-URQXQFDESA-N 0.000 description 2
- 229930182566 Gentamicin Natural products 0.000 description 2
- AEMRFAOFKBGASW-UHFFFAOYSA-N Glycolic acid Chemical compound OCC(O)=O AEMRFAOFKBGASW-UHFFFAOYSA-N 0.000 description 2
- 208000032843 Hemorrhage Diseases 0.000 description 2
- NTYJJOPFIAHURM-UHFFFAOYSA-N Histamine Chemical compound NCCC1=CN=CN1 NTYJJOPFIAHURM-UHFFFAOYSA-N 0.000 description 2
- 108010033040 Histones Proteins 0.000 description 2
- 102000006947 Histones Human genes 0.000 description 2
- 101000944361 Homo sapiens Cyclin-dependent kinase inhibitor 1B Proteins 0.000 description 2
- 101000599951 Homo sapiens Insulin-like growth factor I Proteins 0.000 description 2
- 241000725303 Human immunodeficiency virus Species 0.000 description 2
- 102100037852 Insulin-like growth factor I Human genes 0.000 description 2
- ZDXPYRJPNDTMRX-VKHMYHEASA-N L-glutamine Chemical compound OC(=O)[C@@H](N)CCC(N)=O ZDXPYRJPNDTMRX-VKHMYHEASA-N 0.000 description 2
- 229930182816 L-glutamine Natural products 0.000 description 2
- 208000031422 Lymphocytic Chronic B-Cell Leukemia Diseases 0.000 description 2
- TWRXJAOTZQYOKJ-UHFFFAOYSA-L Magnesium chloride Chemical compound [Mg+2].[Cl-].[Cl-] TWRXJAOTZQYOKJ-UHFFFAOYSA-L 0.000 description 2
- 208000007054 Medullary Carcinoma Diseases 0.000 description 2
- AFVFQIVMOAPDHO-UHFFFAOYSA-N Methanesulfonic acid Chemical compound CS(O)(=O)=O AFVFQIVMOAPDHO-UHFFFAOYSA-N 0.000 description 2
- 208000003445 Mouth Neoplasms Diseases 0.000 description 2
- 241000699660 Mus musculus Species 0.000 description 2
- PXHVJJICTQNCMI-UHFFFAOYSA-N Nickel Chemical compound [Ni] PXHVJJICTQNCMI-UHFFFAOYSA-N 0.000 description 2
- 102000008297 Nuclear Matrix-Associated Proteins Human genes 0.000 description 2
- 108010035916 Nuclear Matrix-Associated Proteins Proteins 0.000 description 2
- 108700020796 Oncogene Proteins 0.000 description 2
- 206010033128 Ovarian cancer Diseases 0.000 description 2
- 206010061902 Pancreatic neoplasm Diseases 0.000 description 2
- 208000007452 Plasmacytoma Diseases 0.000 description 2
- QQONPFPTGQHPMA-UHFFFAOYSA-N Propene Chemical group CC=C QQONPFPTGQHPMA-UHFFFAOYSA-N 0.000 description 2
- 102000007066 Prostate-Specific Antigen Human genes 0.000 description 2
- 108010072866 Prostate-Specific Antigen Proteins 0.000 description 2
- 102000009516 Protein Serine-Threonine Kinases Human genes 0.000 description 2
- 108010009341 Protein Serine-Threonine Kinases Proteins 0.000 description 2
- LCTONWCANYUPML-UHFFFAOYSA-N Pyruvic acid Chemical compound CC(=O)C(O)=O LCTONWCANYUPML-UHFFFAOYSA-N 0.000 description 2
- 208000015634 Rectal Neoplasms Diseases 0.000 description 2
- 229920002684 Sepharose Polymers 0.000 description 2
- VYPSYNLAJGMNEJ-UHFFFAOYSA-N Silicium dioxide Chemical compound O=[Si]=O VYPSYNLAJGMNEJ-UHFFFAOYSA-N 0.000 description 2
- QAOWNCQODCNURD-UHFFFAOYSA-N Sulfuric acid Chemical compound OS(O)(=O)=O QAOWNCQODCNURD-UHFFFAOYSA-N 0.000 description 2
- 206010042971 T-cell lymphoma Diseases 0.000 description 2
- 208000024313 Testicular Neoplasms Diseases 0.000 description 2
- IQFYYKKMVGJFEH-XLPZGREQSA-N Thymidine Chemical compound O=C1NC(=O)C(C)=CN1[C@@H]1O[C@H](CO)[C@@H](O)C1 IQFYYKKMVGJFEH-XLPZGREQSA-N 0.000 description 2
- 102000006288 Transcription Factor TFIIH Human genes 0.000 description 2
- 108010083256 Transcription Factor TFIIH Proteins 0.000 description 2
- 208000007097 Urinary Bladder Neoplasms Diseases 0.000 description 2
- 201000003761 Vaginal carcinoma Diseases 0.000 description 2
- 239000002253 acid Substances 0.000 description 2
- 208000017733 acquired polycythemia vera Diseases 0.000 description 2
- 230000003213 activating effect Effects 0.000 description 2
- 230000004913 activation Effects 0.000 description 2
- 230000001919 adrenal effect Effects 0.000 description 2
- 230000032683 aging Effects 0.000 description 2
- 229930013930 alkaloid Natural products 0.000 description 2
- 150000001412 amines Chemical class 0.000 description 2
- 238000004458 analytical method Methods 0.000 description 2
- 239000000427 antigen Substances 0.000 description 2
- 108091007433 antigens Proteins 0.000 description 2
- 102000036639 antigens Human genes 0.000 description 2
- 239000012062 aqueous buffer Substances 0.000 description 2
- 101150044616 araC gene Proteins 0.000 description 2
- 239000010425 asbestos Substances 0.000 description 2
- 229960003272 asparaginase Drugs 0.000 description 2
- DCXYFEDJOCDNAF-UHFFFAOYSA-M asparaginate Chemical compound [O-]C(=O)C(N)CC(N)=O DCXYFEDJOCDNAF-UHFFFAOYSA-M 0.000 description 2
- 210000003719 b-lymphocyte Anatomy 0.000 description 2
- 230000033228 biological regulation Effects 0.000 description 2
- 210000004556 brain Anatomy 0.000 description 2
- 102000028861 calmodulin binding Human genes 0.000 description 2
- 108091000084 calmodulin binding Proteins 0.000 description 2
- 239000000969 carrier Substances 0.000 description 2
- 230000010261 cell growth Effects 0.000 description 2
- 230000004663 cell proliferation Effects 0.000 description 2
- 230000009134 cell regulation Effects 0.000 description 2
- 210000003169 central nervous system Anatomy 0.000 description 2
- 239000007795 chemical reaction product Substances 0.000 description 2
- 238000004587 chromatography analysis Methods 0.000 description 2
- 208000024207 chronic leukemia Diseases 0.000 description 2
- 208000032852 chronic lymphocytic leukemia Diseases 0.000 description 2
- 230000007882 cirrhosis Effects 0.000 description 2
- 208000019425 cirrhosis of liver Diseases 0.000 description 2
- 229960004316 cisplatin Drugs 0.000 description 2
- DQLATGHUWYMOKM-UHFFFAOYSA-L cisplatin Chemical compound N[Pt](N)(Cl)Cl DQLATGHUWYMOKM-UHFFFAOYSA-L 0.000 description 2
- 210000002808 connective tissue Anatomy 0.000 description 2
- 230000001276 controlling effect Effects 0.000 description 2
- 210000003792 cranial nerve Anatomy 0.000 description 2
- XCIXKGXIYUWCLL-UHFFFAOYSA-N cyclopentanol Chemical compound OC1CCCC1 XCIXKGXIYUWCLL-UHFFFAOYSA-N 0.000 description 2
- 125000001559 cyclopropyl group Chemical group [H]C1([H])C([H])([H])C1([H])* 0.000 description 2
- IGSKHXTUVXSOMB-UHFFFAOYSA-N cyclopropylmethanamine Chemical compound NCC1CC1 IGSKHXTUVXSOMB-UHFFFAOYSA-N 0.000 description 2
- 235000005911 diet Nutrition 0.000 description 2
- 230000037213 diet Effects 0.000 description 2
- FAMRKDQNMBBFBR-BQYQJAHWSA-N diethyl azodicarboxylate Substances CCOC(=O)\N=N\C(=O)OCC FAMRKDQNMBBFBR-BQYQJAHWSA-N 0.000 description 2
- 238000010790 dilution Methods 0.000 description 2
- 239000012895 dilution Substances 0.000 description 2
- 239000000975 dye Substances 0.000 description 2
- 229940088598 enzyme Drugs 0.000 description 2
- 238000001952 enzyme assay Methods 0.000 description 2
- 210000002745 epiphysis Anatomy 0.000 description 2
- 230000000925 erythroid effect Effects 0.000 description 2
- 125000001495 ethyl group Chemical group [H]C([H])([H])C([H])([H])* 0.000 description 2
- FAMRKDQNMBBFBR-UHFFFAOYSA-N ethyl n-ethoxycarbonyliminocarbamate Chemical compound CCOC(=O)N=NC(=O)OCC FAMRKDQNMBBFBR-UHFFFAOYSA-N 0.000 description 2
- 238000002474 experimental method Methods 0.000 description 2
- 210000000744 eyelid Anatomy 0.000 description 2
- 239000012894 fetal calf serum Substances 0.000 description 2
- 201000004098 fibrolamellar carcinoma Diseases 0.000 description 2
- 238000001914 filtration Methods 0.000 description 2
- 229960002074 flutamide Drugs 0.000 description 2
- MKXKFYHWDHIYRV-UHFFFAOYSA-N flutamide Chemical compound CC(C)C(=O)NC1=CC=C([N+]([O-])=O)C(C(F)(F)F)=C1 MKXKFYHWDHIYRV-UHFFFAOYSA-N 0.000 description 2
- 235000011389 fruit/vegetable juice Nutrition 0.000 description 2
- 206010017758 gastric cancer Diseases 0.000 description 2
- 210000004392 genitalia Anatomy 0.000 description 2
- 230000000762 glandular Effects 0.000 description 2
- RWSXRVCMGQZWBV-WDSKDSINSA-N glutathione Chemical compound OC(=O)[C@@H](N)CCC(=O)N[C@@H](CS)C(=O)NCC(O)=O RWSXRVCMGQZWBV-WDSKDSINSA-N 0.000 description 2
- 210000002288 golgi apparatus Anatomy 0.000 description 2
- 239000001963 growth medium Substances 0.000 description 2
- 208000014829 head and neck neoplasm Diseases 0.000 description 2
- 210000003958 hematopoietic stem cell Anatomy 0.000 description 2
- 230000002440 hepatic effect Effects 0.000 description 2
- 206010073071 hepatocellular carcinoma Diseases 0.000 description 2
- 231100000844 hepatocellular carcinoma Toxicity 0.000 description 2
- 238000002513 implantation Methods 0.000 description 2
- 201000004933 in situ carcinoma Diseases 0.000 description 2
- 108091006086 inhibitor proteins Proteins 0.000 description 2
- 229940079322 interferon Drugs 0.000 description 2
- 229940047124 interferons Drugs 0.000 description 2
- SUMDYPCJJOFFON-UHFFFAOYSA-N isethionic acid Chemical compound OCCS(O)(=O)=O SUMDYPCJJOFFON-UHFFFAOYSA-N 0.000 description 2
- 125000000959 isobutyl group Chemical group [H]C([H])([H])C([H])(C([H])([H])[H])C([H])([H])* 0.000 description 2
- 230000002147 killing effect Effects 0.000 description 2
- JVTAAEKCZFNVCJ-UHFFFAOYSA-N lactic acid Chemical compound CC(O)C(O)=O JVTAAEKCZFNVCJ-UHFFFAOYSA-N 0.000 description 2
- 239000011344 liquid material Substances 0.000 description 2
- 239000007937 lozenge Substances 0.000 description 2
- 210000004698 lymphocyte Anatomy 0.000 description 2
- 239000006166 lysate Substances 0.000 description 2
- 239000000463 material Substances 0.000 description 2
- 208000023356 medullary thyroid gland carcinoma Diseases 0.000 description 2
- GLVAUDGFNGKCSF-UHFFFAOYSA-N mercaptopurine Chemical compound S=C1NC=NC2=C1NC=N2 GLVAUDGFNGKCSF-UHFFFAOYSA-N 0.000 description 2
- 108020004999 messenger RNA Proteins 0.000 description 2
- 125000002496 methyl group Chemical group [H]C([H])([H])* 0.000 description 2
- 210000003632 microfilament Anatomy 0.000 description 2
- 150000007522 mineralic acids Chemical class 0.000 description 2
- 210000000479 mitotic spindle apparatus Anatomy 0.000 description 2
- 125000004108 n-butyl group Chemical group [H]C([H])([H])C([H])([H])C([H])([H])C([H])([H])* 0.000 description 2
- 230000009826 neoplastic cell growth Effects 0.000 description 2
- 230000009223 neuronal apoptosis Effects 0.000 description 2
- 230000003961 neuronal insult Effects 0.000 description 2
- 210000002353 nuclear lamina Anatomy 0.000 description 2
- 210000004940 nucleus Anatomy 0.000 description 2
- 239000008203 oral pharmaceutical composition Substances 0.000 description 2
- 150000007524 organic acids Chemical class 0.000 description 2
- 239000003960 organic solvent Substances 0.000 description 2
- 229960003552 other antineoplastic agent in atc Drugs 0.000 description 2
- 238000012261 overproduction Methods 0.000 description 2
- DPBLXKKOBLCELK-UHFFFAOYSA-N pentan-1-amine Chemical compound CCCCCN DPBLXKKOBLCELK-UHFFFAOYSA-N 0.000 description 2
- YBYRMVIVWMBXKQ-UHFFFAOYSA-N phenylmethanesulfonyl fluoride Chemical compound FS(=O)(=O)CC1=CC=CC=C1 YBYRMVIVWMBXKQ-UHFFFAOYSA-N 0.000 description 2
- 210000003635 pituitary gland Anatomy 0.000 description 2
- 230000000750 progressive effect Effects 0.000 description 2
- WGYKZJWCGVVSQN-UHFFFAOYSA-N propylamine Chemical compound CCCN WGYKZJWCGVVSQN-UHFFFAOYSA-N 0.000 description 2
- 239000000649 purine antagonist Substances 0.000 description 2
- KIDHWZJUCRJVML-UHFFFAOYSA-N putrescine Chemical compound NCCCCN KIDHWZJUCRJVML-UHFFFAOYSA-N 0.000 description 2
- 239000003790 pyrimidine antagonist Substances 0.000 description 2
- 102000005962 receptors Human genes 0.000 description 2
- 108020003175 receptors Proteins 0.000 description 2
- 230000002829 reductive effect Effects 0.000 description 2
- 238000010992 reflux Methods 0.000 description 2
- 229910052895 riebeckite Inorganic materials 0.000 description 2
- 102200127349 rs11547328 Human genes 0.000 description 2
- 125000002914 sec-butyl group Chemical group [H]C([H])([H])C([H])([H])C([H])(*)C([H])([H])[H] 0.000 description 2
- 239000012056 semi-solid material Substances 0.000 description 2
- QZAYGJVTTNCVMB-UHFFFAOYSA-N serotonin Chemical compound C1=C(O)C=C2C(CCN)=CNC2=C1 QZAYGJVTTNCVMB-UHFFFAOYSA-N 0.000 description 2
- 239000000741 silica gel Substances 0.000 description 2
- 229910002027 silica gel Inorganic materials 0.000 description 2
- 210000003625 skull Anatomy 0.000 description 2
- 230000000391 smoking effect Effects 0.000 description 2
- 239000011780 sodium chloride Substances 0.000 description 2
- 239000011343 solid material Substances 0.000 description 2
- 239000000126 substance Substances 0.000 description 2
- 239000000829 suppository Substances 0.000 description 2
- 239000000725 suspension Substances 0.000 description 2
- 208000024891 symptom Diseases 0.000 description 2
- 239000003826 tablet Substances 0.000 description 2
- 125000000999 tert-butyl group Chemical group [H]C([H])([H])C(*)(C([H])([H])[H])C([H])([H])[H] 0.000 description 2
- 230000001573 trophoblastic effect Effects 0.000 description 2
- APJYDQYYACXCRM-UHFFFAOYSA-N tryptamine Chemical compound C1=CC=C2C(CCN)=CNC2=C1 APJYDQYYACXCRM-UHFFFAOYSA-N 0.000 description 2
- 208000029729 tumor suppressor gene on chromosome 11 Diseases 0.000 description 2
- 241000701447 unidentified baculovirus Species 0.000 description 2
- 201000005112 urinary bladder cancer Diseases 0.000 description 2
- RQEUFEKYXDPUSK-SSDOTTSWSA-N (1R)-1-phenylethanamine Chemical compound C[C@@H](N)C1=CC=CC=C1 RQEUFEKYXDPUSK-SSDOTTSWSA-N 0.000 description 1
- RQEUFEKYXDPUSK-ZETCQYMHSA-N (1S)-1-phenylethanamine Chemical compound C[C@H](N)C1=CC=CC=C1 RQEUFEKYXDPUSK-ZETCQYMHSA-N 0.000 description 1
- SJUKJZSTBBSGHF-UHFFFAOYSA-N (2,4-dichlorophenyl)methanamine Chemical compound NCC1=CC=C(Cl)C=C1Cl SJUKJZSTBBSGHF-UHFFFAOYSA-N 0.000 description 1
- KDDNKZCVYQDGKE-UHFFFAOYSA-N (2-chlorophenyl)methanamine Chemical compound NCC1=CC=CC=C1Cl KDDNKZCVYQDGKE-UHFFFAOYSA-N 0.000 description 1
- LRFWYBZWRQWZIM-UHFFFAOYSA-N (2-fluorophenyl)methanamine Chemical compound NCC1=CC=CC=C1F LRFWYBZWRQWZIM-UHFFFAOYSA-N 0.000 description 1
- IXHNFOOSLAWRBQ-UHFFFAOYSA-N (3,4-dichlorophenyl)methanamine Chemical compound NCC1=CC=C(Cl)C(Cl)=C1 IXHNFOOSLAWRBQ-UHFFFAOYSA-N 0.000 description 1
- LQLOGZQVKUNBRX-UHFFFAOYSA-N (3-iodophenyl)methanamine Chemical compound NCC1=CC=CC(I)=C1 LQLOGZQVKUNBRX-UHFFFAOYSA-N 0.000 description 1
- RGXUCUWVGKLACF-UHFFFAOYSA-N (3-methylphenyl)methanamine Chemical compound CC1=CC=CC(CN)=C1 RGXUCUWVGKLACF-UHFFFAOYSA-N 0.000 description 1
- ODMMHGMIZYLHNN-UHFFFAOYSA-N (4-chloro-2-fluorophenyl)methanamine Chemical compound NCC1=CC=C(Cl)C=C1F ODMMHGMIZYLHNN-UHFFFAOYSA-N 0.000 description 1
- YMVFJGSXZNNUDW-UHFFFAOYSA-N (4-chlorophenyl)methanamine Chemical compound NCC1=CC=C(Cl)C=C1 YMVFJGSXZNNUDW-UHFFFAOYSA-N 0.000 description 1
- IIFVWLUQBAIPMJ-UHFFFAOYSA-N (4-fluorophenyl)methanamine Chemical compound NCC1=CC=C(F)C=C1 IIFVWLUQBAIPMJ-UHFFFAOYSA-N 0.000 description 1
- PWGJDPKCLMLPJW-UHFFFAOYSA-N 1,8-diaminooctane Chemical compound NCCCCCCCCN PWGJDPKCLMLPJW-UHFFFAOYSA-N 0.000 description 1
- VXNZUUAINFGPBY-UHFFFAOYSA-N 1-Butene Chemical group CCC=C VXNZUUAINFGPBY-UHFFFAOYSA-N 0.000 description 1
- BMVXCPBXGZKUPN-UHFFFAOYSA-N 1-hexanamine Chemical compound CCCCCCN BMVXCPBXGZKUPN-UHFFFAOYSA-N 0.000 description 1
- OPMFFAOEPFATTG-UHFFFAOYSA-N 2,2,2-trifluoroethylhydrazine Chemical compound NNCC(F)(F)F OPMFFAOEPFATTG-UHFFFAOYSA-N 0.000 description 1
- GXVUZYLYWKWJIM-UHFFFAOYSA-N 2-(2-aminoethoxy)ethanamine Chemical compound NCCOCCN GXVUZYLYWKWJIM-UHFFFAOYSA-N 0.000 description 1
- DHPOUEPMENIWGX-UHFFFAOYSA-N 2-(aminomethyl)butan-1-ol Chemical compound CCC(CN)CO DHPOUEPMENIWGX-UHFFFAOYSA-N 0.000 description 1
- IOOMXAQUNPWDLL-UHFFFAOYSA-N 2-[6-(diethylamino)-3-(diethyliminiumyl)-3h-xanthen-9-yl]-5-sulfobenzene-1-sulfonate Chemical compound C=12C=CC(=[N+](CC)CC)C=C2OC2=CC(N(CC)CC)=CC=C2C=1C1=CC=C(S(O)(=O)=O)C=C1S([O-])(=O)=O IOOMXAQUNPWDLL-UHFFFAOYSA-N 0.000 description 1
- WOQKMUDQRKXVLO-UHFFFAOYSA-N 2-[[7-methyl-6-[(phenylmethyl)amino]-2-purinyl]amino]ethanol Chemical compound C=12N(C)C=NC2=NC(NCCO)=NC=1NCC1=CC=CC=C1 WOQKMUDQRKXVLO-UHFFFAOYSA-N 0.000 description 1
- 125000004847 2-fluorobenzyl group Chemical group [H]C1=C([H])C(F)=C(C([H])=C1[H])C([H])([H])* 0.000 description 1
- GBHCABUWWQUMAJ-UHFFFAOYSA-N 2-hydrazinoethanol Chemical compound NNCCO GBHCABUWWQUMAJ-UHFFFAOYSA-N 0.000 description 1
- ZOOGRGPOEVQQDX-UUOKFMHZSA-N 3',5'-cyclic GMP Chemical compound C([C@H]1O2)OP(O)(=O)O[C@H]1[C@@H](O)[C@@H]2N1C(N=C(NC2=O)N)=C2N=C1 ZOOGRGPOEVQQDX-UUOKFMHZSA-N 0.000 description 1
- HQNOODJDSFSURF-UHFFFAOYSA-N 3-(1h-imidazol-2-yl)propan-1-amine Chemical compound NCCCC1=NC=CN1 HQNOODJDSFSURF-UHFFFAOYSA-N 0.000 description 1
- BMYNFMYTOJXKLE-UHFFFAOYSA-N 3-azaniumyl-2-hydroxypropanoate Chemical compound NCC(O)C(O)=O BMYNFMYTOJXKLE-UHFFFAOYSA-N 0.000 description 1
- KDHWOCLBMVSZPG-UHFFFAOYSA-N 3-imidazol-1-ylpropan-1-amine Chemical compound NCCCN1C=CN=C1 KDHWOCLBMVSZPG-UHFFFAOYSA-N 0.000 description 1
- UIKUBYKUYUSRSM-UHFFFAOYSA-N 3-morpholinopropylamine Chemical compound NCCCN1CCOCC1 UIKUBYKUYUSRSM-UHFFFAOYSA-N 0.000 description 1
- LYUQWQRTDLVQGA-UHFFFAOYSA-N 3-phenylpropylamine Chemical compound NCCCC1=CC=CC=C1 LYUQWQRTDLVQGA-UHFFFAOYSA-N 0.000 description 1
- QFVHZQCOUORWEI-UHFFFAOYSA-N 4-[(4-anilino-5-sulfonaphthalen-1-yl)diazenyl]-5-hydroxynaphthalene-2,7-disulfonic acid Chemical compound C=12C(O)=CC(S(O)(=O)=O)=CC2=CC(S(O)(=O)=O)=CC=1N=NC(C1=CC=CC(=C11)S(O)(=O)=O)=CC=C1NC1=CC=CC=C1 QFVHZQCOUORWEI-UHFFFAOYSA-N 0.000 description 1
- QCQCHGYLTSGIGX-GHXANHINSA-N 4-[[(3ar,5ar,5br,7ar,9s,11ar,11br,13as)-5a,5b,8,8,11a-pentamethyl-3a-[(5-methylpyridine-3-carbonyl)amino]-2-oxo-1-propan-2-yl-4,5,6,7,7a,9,10,11,11b,12,13,13a-dodecahydro-3h-cyclopenta[a]chrysen-9-yl]oxy]-2,2-dimethyl-4-oxobutanoic acid Chemical compound N([C@@]12CC[C@@]3(C)[C@]4(C)CC[C@H]5C(C)(C)[C@@H](OC(=O)CC(C)(C)C(O)=O)CC[C@]5(C)[C@H]4CC[C@@H]3C1=C(C(C2)=O)C(C)C)C(=O)C1=CN=CC(C)=C1 QCQCHGYLTSGIGX-GHXANHINSA-N 0.000 description 1
- AGNFWIZBEATIAK-UHFFFAOYSA-N 4-phenylbutylamine Chemical compound NCCCCC1=CC=CC=C1 AGNFWIZBEATIAK-UHFFFAOYSA-N 0.000 description 1
- LQGKDMHENBFVRC-UHFFFAOYSA-N 5-aminopentan-1-ol Chemical compound NCCCCCO LQGKDMHENBFVRC-UHFFFAOYSA-N 0.000 description 1
- JZNBMCOSOXIZJB-UHFFFAOYSA-N 6-amino-2-methylheptan-2-ol;hydron;chloride Chemical compound Cl.CC(N)CCCC(C)(C)O JZNBMCOSOXIZJB-UHFFFAOYSA-N 0.000 description 1
- STQGQHZAVUOBTE-UHFFFAOYSA-N 7-Cyan-hept-2t-en-4,6-diinsaeure Natural products C1=2C(O)=C3C(=O)C=4C(OC)=CC=CC=4C(=O)C3=C(O)C=2CC(O)(C(C)=O)CC1OC1CC(N)C(O)C(C)O1 STQGQHZAVUOBTE-UHFFFAOYSA-N 0.000 description 1
- ZCYVEMRRCGMTRW-UHFFFAOYSA-N 7553-56-2 Chemical group [I] ZCYVEMRRCGMTRW-UHFFFAOYSA-N 0.000 description 1
- 208000004998 Abdominal Pain Diseases 0.000 description 1
- 108010043137 Actomyosin Proteins 0.000 description 1
- 241000321096 Adenoides Species 0.000 description 1
- 208000003200 Adenoma Diseases 0.000 description 1
- ZHGNHOOVYPHPNJ-UHFFFAOYSA-N Amigdalin Chemical compound FC(F)(F)C(=O)OCC1OC(OCC2OC(OC(C#N)C3=CC=CC=C3)C(OC(=O)C(F)(F)F)C(OC(=O)C(F)(F)F)C2OC(=O)C(F)(F)F)C(OC(=O)C(F)(F)F)C(OC(=O)C(F)(F)F)C1OC(=O)C(F)(F)F ZHGNHOOVYPHPNJ-UHFFFAOYSA-N 0.000 description 1
- 244000144725 Amygdalus communis Species 0.000 description 1
- 102100032187 Androgen receptor Human genes 0.000 description 1
- 102000008102 Ankyrins Human genes 0.000 description 1
- 108010049777 Ankyrins Proteins 0.000 description 1
- 208000007860 Anus Neoplasms Diseases 0.000 description 1
- 108010039627 Aprotinin Proteins 0.000 description 1
- 208000022211 Arteriovenous Malformations Diseases 0.000 description 1
- DCXYFEDJOCDNAF-UHFFFAOYSA-N Asparagine Natural products OC(=O)C(N)CC(N)=O DCXYFEDJOCDNAF-UHFFFAOYSA-N 0.000 description 1
- 208000003950 B-cell lymphoma Diseases 0.000 description 1
- 108700020463 BRCA1 Proteins 0.000 description 1
- 102000036365 BRCA1 Human genes 0.000 description 1
- 241000894006 Bacteria Species 0.000 description 1
- 208000029856 Bartholin gland adenocarcinoma Diseases 0.000 description 1
- 108700004676 Bence Jones Proteins 0.000 description 1
- DWRXFEITVBNRMK-UHFFFAOYSA-N Beta-D-1-Arabinofuranosylthymine Natural products O=C1NC(=O)C(C)=CN1C1C(O)C(O)C(CO)O1 DWRXFEITVBNRMK-UHFFFAOYSA-N 0.000 description 1
- 108010006654 Bleomycin Proteins 0.000 description 1
- 206010073106 Bone giant cell tumour malignant Diseases 0.000 description 1
- 208000018084 Bone neoplasm Diseases 0.000 description 1
- 101800004538 Bradykinin Proteins 0.000 description 1
- 208000003174 Brain Neoplasms Diseases 0.000 description 1
- WKBOTKDWSSQWDR-UHFFFAOYSA-N Bromine atom Chemical compound [Br] WKBOTKDWSSQWDR-UHFFFAOYSA-N 0.000 description 1
- 101150025841 CCND1 gene Proteins 0.000 description 1
- 108010034798 CDC2 Protein Kinase Proteins 0.000 description 1
- 102000009728 CDC2 Protein Kinase Human genes 0.000 description 1
- 101000909256 Caldicellulosiruptor bescii (strain ATCC BAA-1888 / DSM 6725 / Z-1320) DNA polymerase I Proteins 0.000 description 1
- 102000000584 Calmodulin Human genes 0.000 description 1
- 108010041952 Calmodulin Proteins 0.000 description 1
- OKTJSMMVPCPJKN-UHFFFAOYSA-N Carbon Chemical compound [C] OKTJSMMVPCPJKN-UHFFFAOYSA-N 0.000 description 1
- DLGOEMSEDOSKAD-UHFFFAOYSA-N Carmustine Chemical compound ClCCNC(=O)N(N=O)CCCl DLGOEMSEDOSKAD-UHFFFAOYSA-N 0.000 description 1
- 101150053721 Cdk5 gene Proteins 0.000 description 1
- 101150090188 Cdk8 gene Proteins 0.000 description 1
- 108010059892 Cellulase Proteins 0.000 description 1
- ZAMOUSCENKQFHK-UHFFFAOYSA-N Chlorine atom Chemical compound [Cl] ZAMOUSCENKQFHK-UHFFFAOYSA-N 0.000 description 1
- 201000005262 Chondroma Diseases 0.000 description 1
- 201000009047 Chordoma Diseases 0.000 description 1
- 208000017667 Chronic Disease Diseases 0.000 description 1
- CGOKCCVAARDEHZ-GJFNAKTISA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCCc2ccccn2)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCCc2ccccn2)c2ncn(C3CCCC3)c2n1 CGOKCCVAARDEHZ-GJFNAKTISA-N 0.000 description 1
- BRIDXNWDYVJTIN-VAJGKAQFSA-N Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2ccccc2F)c2ncn(C3CCCC3)c2n1 Chemical compound Cl.Cl.N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2ccccc2F)c2ncn(C3CCCC3)c2n1 BRIDXNWDYVJTIN-VAJGKAQFSA-N 0.000 description 1
- 208000002881 Colic Diseases 0.000 description 1
- 206010048832 Colon adenoma Diseases 0.000 description 1
- 208000001333 Colorectal Neoplasms Diseases 0.000 description 1
- 206010053138 Congenital aplastic anaemia Diseases 0.000 description 1
- 208000009798 Craniopharyngioma Diseases 0.000 description 1
- 206010011703 Cyanosis Diseases 0.000 description 1
- 102000002428 Cyclin C Human genes 0.000 description 1
- 108010068155 Cyclin C Proteins 0.000 description 1
- 108010058544 Cyclin D2 Proteins 0.000 description 1
- 108010058545 Cyclin D3 Proteins 0.000 description 1
- 108010009392 Cyclin-Dependent Kinase Inhibitor p16 Proteins 0.000 description 1
- 102100026810 Cyclin-dependent kinase 7 Human genes 0.000 description 1
- 101710106276 Cyclin-dependent kinase 7 Proteins 0.000 description 1
- 102100033270 Cyclin-dependent kinase inhibitor 1 Human genes 0.000 description 1
- GVOUFPWUYJWQSK-UHFFFAOYSA-N Cyclofenil Chemical group C1=CC(OC(=O)C)=CC=C1C(C=1C=CC(OC(C)=O)=CC=1)=C1CCCCC1 GVOUFPWUYJWQSK-UHFFFAOYSA-N 0.000 description 1
- CMSMOCZEIVJLDB-UHFFFAOYSA-N Cyclophosphamide Chemical compound ClCCN(CCCl)P1(=O)NCCCO1 CMSMOCZEIVJLDB-UHFFFAOYSA-N 0.000 description 1
- UHDGCWIWMRVCDJ-CCXZUQQUSA-N Cytarabine Chemical compound O=C1N=C(N)C=CN1[C@H]1[C@@H](O)[C@H](O)[C@@H](CO)O1 UHDGCWIWMRVCDJ-CCXZUQQUSA-N 0.000 description 1
- 108010017826 DNA Polymerase I Proteins 0.000 description 1
- 102000004594 DNA Polymerase I Human genes 0.000 description 1
- 102000007528 DNA Polymerase III Human genes 0.000 description 1
- 108010071146 DNA Polymerase III Proteins 0.000 description 1
- 230000004544 DNA amplification Effects 0.000 description 1
- 108050008316 DNA endonuclease RBBP8 Proteins 0.000 description 1
- 101710177611 DNA polymerase II large subunit Proteins 0.000 description 1
- 101710184669 DNA polymerase II small subunit Proteins 0.000 description 1
- 108010014303 DNA-directed DNA polymerase Proteins 0.000 description 1
- 102000016928 DNA-directed DNA polymerase Human genes 0.000 description 1
- 102000004163 DNA-directed RNA polymerases Human genes 0.000 description 1
- 108090000626 DNA-directed RNA polymerases Proteins 0.000 description 1
- 101100239628 Danio rerio myca gene Proteins 0.000 description 1
- WEAHRLBPCANXCN-UHFFFAOYSA-N Daunomycin Natural products CCC1(O)CC(OC2CC(N)C(O)C(C)O2)c3cc4C(=O)c5c(OC)cccc5C(=O)c4c(O)c3C1 WEAHRLBPCANXCN-UHFFFAOYSA-N 0.000 description 1
- 208000001154 Dermoid Cyst Diseases 0.000 description 1
- 102100036912 Desmin Human genes 0.000 description 1
- 108010044052 Desmin Proteins 0.000 description 1
- 201000010374 Down Syndrome Diseases 0.000 description 1
- 108050002772 E3 ubiquitin-protein ligase Mdm2 Proteins 0.000 description 1
- 102000012199 E3 ubiquitin-protein ligase Mdm2 Human genes 0.000 description 1
- 102000002322 Egg Proteins Human genes 0.000 description 1
- 108010000912 Egg Proteins Proteins 0.000 description 1
- 241000196324 Embryophyta Species 0.000 description 1
- 208000001976 Endocrine Gland Neoplasms Diseases 0.000 description 1
- 206010014967 Ependymoma Diseases 0.000 description 1
- 208000031637 Erythroblastic Acute Leukemia Diseases 0.000 description 1
- 208000036566 Erythroleukaemia Diseases 0.000 description 1
- 241000588724 Escherichia coli Species 0.000 description 1
- 206010073306 Exposure to radiation Diseases 0.000 description 1
- 208000005163 Extra-Adrenal Paraganglioma Diseases 0.000 description 1
- 208000031206 Familial polycythaemia Diseases 0.000 description 1
- 201000004939 Fanconi anemia Diseases 0.000 description 1
- PXGOKWXKJXAPGV-UHFFFAOYSA-N Fluorine Chemical compound FF PXGOKWXKJXAPGV-UHFFFAOYSA-N 0.000 description 1
- GHASVSINZRGABV-UHFFFAOYSA-N Fluorouracil Chemical compound FC1=CNC(=O)NC1=O GHASVSINZRGABV-UHFFFAOYSA-N 0.000 description 1
- 206010017533 Fungal infection Diseases 0.000 description 1
- 102100024185 G1/S-specific cyclin-D2 Human genes 0.000 description 1
- 102100037859 G1/S-specific cyclin-D3 Human genes 0.000 description 1
- 241000287828 Gallus gallus Species 0.000 description 1
- 102400000921 Gastrin Human genes 0.000 description 1
- 108010052343 Gastrins Proteins 0.000 description 1
- 230000010558 Gene Alterations Effects 0.000 description 1
- 208000034826 Genetic Predisposition to Disease Diseases 0.000 description 1
- 208000007569 Giant Cell Tumors Diseases 0.000 description 1
- 108010024636 Glutathione Proteins 0.000 description 1
- 206010018691 Granuloma Diseases 0.000 description 1
- QXZGBUJJYSLZLT-UHFFFAOYSA-N H-Arg-Pro-Pro-Gly-Phe-Ser-Pro-Phe-Arg-OH Natural products NC(N)=NCCCC(N)C(=O)N1CCCC1C(=O)N1C(C(=O)NCC(=O)NC(CC=2C=CC=CC=2)C(=O)NC(CO)C(=O)N2C(CCC2)C(=O)NC(CC=2C=CC=CC=2)C(=O)NC(CCCN=C(N)N)C(O)=O)CCC1 QXZGBUJJYSLZLT-UHFFFAOYSA-N 0.000 description 1
- 208000002927 Hamartoma Diseases 0.000 description 1
- 206010019629 Hepatic adenoma Diseases 0.000 description 1
- 241000700721 Hepatitis B virus Species 0.000 description 1
- 241000282412 Homo Species 0.000 description 1
- 101000980920 Homo sapiens Cyclin-dependent kinase 4 inhibitor D Proteins 0.000 description 1
- 101000944380 Homo sapiens Cyclin-dependent kinase inhibitor 1 Proteins 0.000 description 1
- 101000980756 Homo sapiens G1/S-specific cyclin-D1 Proteins 0.000 description 1
- 101001072338 Homo sapiens Proliferating cell nuclear antigen Proteins 0.000 description 1
- 241000598436 Human T-cell lymphotropic virus Species 0.000 description 1
- 241000701806 Human papillomavirus Species 0.000 description 1
- 206010060377 Hypergastrinaemia Diseases 0.000 description 1
- 201000002980 Hyperparathyroidism Diseases 0.000 description 1
- 206010062767 Hypophysitis Diseases 0.000 description 1
- 108060003951 Immunoglobulin Proteins 0.000 description 1
- 208000005045 Interdigitating dendritic cell sarcoma Diseases 0.000 description 1
- 102000003996 Interferon-beta Human genes 0.000 description 1
- 108090000467 Interferon-beta Proteins 0.000 description 1
- 208000005016 Intestinal Neoplasms Diseases 0.000 description 1
- 208000009164 Islet Cell Adenoma Diseases 0.000 description 1
- VQTUBCCKSQIDNK-UHFFFAOYSA-N Isobutene Chemical group CC(C)=C VQTUBCCKSQIDNK-UHFFFAOYSA-N 0.000 description 1
- 241000701460 JC polyomavirus Species 0.000 description 1
- 208000008839 Kidney Neoplasms Diseases 0.000 description 1
- 102100035792 Kininogen-1 Human genes 0.000 description 1
- DCXYFEDJOCDNAF-REOHCLBHSA-N L-asparagine Chemical compound OC(=O)[C@@H](N)CC(N)=O DCXYFEDJOCDNAF-REOHCLBHSA-N 0.000 description 1
- FBOZXECLQNJBKD-ZDUSSCGKSA-N L-methotrexate Chemical compound C=1N=C2N=C(N)N=C(N)C2=NC=1CN(C)C1=CC=C(C(=O)N[C@@H](CCC(O)=O)C(O)=O)C=C1 FBOZXECLQNJBKD-ZDUSSCGKSA-N 0.000 description 1
- 208000018142 Leiomyosarcoma Diseases 0.000 description 1
- GDBQQVLCIARPGH-UHFFFAOYSA-N Leupeptin Natural products CC(C)CC(NC(C)=O)C(=O)NC(CC(C)C)C(=O)NC(C=O)CCCN=C(N)N GDBQQVLCIARPGH-UHFFFAOYSA-N 0.000 description 1
- 208000002404 Liver Cell Adenoma Diseases 0.000 description 1
- GQYIWUVLTXOXAJ-UHFFFAOYSA-N Lomustine Chemical compound ClCCN(N=O)C(=O)NC1CCCCC1 GQYIWUVLTXOXAJ-UHFFFAOYSA-N 0.000 description 1
- 208000030289 Lymphoproliferative disease Diseases 0.000 description 1
- 101150011786 MKK3 gene Proteins 0.000 description 1
- 101150078498 MYB gene Proteins 0.000 description 1
- 101150039798 MYC gene Proteins 0.000 description 1
- 208000006644 Malignant Fibrous Histiocytoma Diseases 0.000 description 1
- 241000124008 Mammalia Species 0.000 description 1
- 108010075942 Maturation-Promoting Factor Proteins 0.000 description 1
- 102000011961 Maturation-Promoting Factor Human genes 0.000 description 1
- 201000009574 Mesenchymal Chondrosarcoma Diseases 0.000 description 1
- 229930192392 Mitomycin Natural products 0.000 description 1
- 241000700560 Molluscum contagiosum virus Species 0.000 description 1
- 206010060880 Monoclonal gammopathy Diseases 0.000 description 1
- 208000006876 Multiple Endocrine Neoplasia Type 2b Diseases 0.000 description 1
- 206010028193 Multiple endocrine neoplasia syndromes Diseases 0.000 description 1
- 206010073148 Multiple endocrine neoplasia type 2A Diseases 0.000 description 1
- 101000983859 Mycobacterium tuberculosis (strain ATCC 25618 / H37Rv) Lipoprotein LpqH Proteins 0.000 description 1
- 208000033833 Myelomonocytic Chronic Leukemia Diseases 0.000 description 1
- 102000005640 Myosin Type II Human genes 0.000 description 1
- 108010045128 Myosin Type II Proteins 0.000 description 1
- NWIBSHFKIJFRCO-WUDYKRTCSA-N Mytomycin Chemical compound C1N2C(C(C(C)=C(N)C3=O)=O)=C3[C@@H](COC(N)=O)[C@@]2(OC)[C@@H]2[C@H]1N2 NWIBSHFKIJFRCO-WUDYKRTCSA-N 0.000 description 1
- FMLJGXGCEMIFBM-SHTZXODSSA-N NCCCCNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 Chemical compound NCCCCNc1nc(N[C@H]2CC[C@H](N)CC2)nc2n(cnc12)C1CCCC1 FMLJGXGCEMIFBM-SHTZXODSSA-N 0.000 description 1
- FCCOSIPXVGNASY-QAQDUYKDSA-N N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)N(CC)C1=CC=NC=C1 Chemical compound N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)N(CC)C1=CC=NC=C1 FCCOSIPXVGNASY-QAQDUYKDSA-N 0.000 description 1
- SNXXRRMDDFGHBJ-ZNUUHSPMSA-N N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NC(CCCC(C)(C)O)C Chemical compound N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NC(CCCC(C)(C)O)C SNXXRRMDDFGHBJ-ZNUUHSPMSA-N 0.000 description 1
- PMPAKRCPRUPOET-HAQNSBGRSA-N N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC(F)(F)F Chemical compound N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC(F)(F)F PMPAKRCPRUPOET-HAQNSBGRSA-N 0.000 description 1
- ROZHRKXMXFBFOF-QAQDUYKDSA-N N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC1=C(C=C(C=C1)Cl)Cl Chemical compound N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC1=C(C=C(C=C1)Cl)Cl ROZHRKXMXFBFOF-QAQDUYKDSA-N 0.000 description 1
- VWDKLDNZNGSNAY-QAQDUYKDSA-N N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC1=C(C=C(C=C1)Cl)F Chemical compound N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC1=C(C=C(C=C1)Cl)F VWDKLDNZNGSNAY-QAQDUYKDSA-N 0.000 description 1
- LJTAAGRXFNBGJM-QAQDUYKDSA-N N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC1=C(C=CC=C1)Cl Chemical compound N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC1=C(C=CC=C1)Cl LJTAAGRXFNBGJM-QAQDUYKDSA-N 0.000 description 1
- BUZWGLJHSKVXDX-WGSAOQKQSA-N N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC1=CC(=CC=C1)C Chemical compound N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC1=CC(=CC=C1)C BUZWGLJHSKVXDX-WGSAOQKQSA-N 0.000 description 1
- BPUOYTASPPQWKL-IYARVYRRSA-N N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC1=CC(=CC=C1)Cl Chemical compound N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC1=CC(=CC=C1)Cl BPUOYTASPPQWKL-IYARVYRRSA-N 0.000 description 1
- PSGCVPJBHFOIDD-IBOPQAGSSA-N N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC1OCCCC1 Chemical compound N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC1OCCCC1 PSGCVPJBHFOIDD-IBOPQAGSSA-N 0.000 description 1
- LEWKKWQMYKLGBE-WKILWMFISA-N N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC=1NC2=C(N1)C=CC=C2 Chemical compound N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCC=1NC2=C(N1)C=CC=C2 LEWKKWQMYKLGBE-WKILWMFISA-N 0.000 description 1
- FIMZCHIOEVYYRW-WKILWMFISA-N N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCCC(C)C Chemical compound N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCCC(C)C FIMZCHIOEVYYRW-WKILWMFISA-N 0.000 description 1
- MIJAMEAZWKZWKB-IYARVYRRSA-N N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCCC1=CC=C(C=C1)O Chemical compound N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCCC1=CC=C(C=C1)O MIJAMEAZWKZWKB-IYARVYRRSA-N 0.000 description 1
- AQLSHYOBJVRGPU-SAABIXHNSA-N N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCCC1=NC=CC=C1 Chemical compound N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCCC1=NC=CC=C1 AQLSHYOBJVRGPU-SAABIXHNSA-N 0.000 description 1
- XXYNJTYWDKRNGL-MXVIHJGJSA-N N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCCCC1=CC=CC=C1 Chemical compound N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCCCC1=CC=CC=C1 XXYNJTYWDKRNGL-MXVIHJGJSA-N 0.000 description 1
- XYXWQIAHQHODJV-WGSAOQKQSA-N N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCCCCCCCCN Chemical compound N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCCCCCCCCN XYXWQIAHQHODJV-WGSAOQKQSA-N 0.000 description 1
- DOWCLVYMLVVSLN-QAQDUYKDSA-N N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCCCN1C=NC=C1 Chemical compound N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCCCN1C=NC=C1 DOWCLVYMLVVSLN-QAQDUYKDSA-N 0.000 description 1
- RDBGZPXFTYINIO-IYARVYRRSA-N N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCCCN1CCOCC1 Chemical compound N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NCCCN1CCOCC1 RDBGZPXFTYINIO-IYARVYRRSA-N 0.000 description 1
- YTCYSJNVQOCFMF-HAQNSBGRSA-N N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NNCC(F)(F)F Chemical compound N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NNCC(F)(F)F YTCYSJNVQOCFMF-HAQNSBGRSA-N 0.000 description 1
- JYEDZKFFPWKMOJ-JOCQHMNTSA-N N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NNCCO Chemical compound N[C@@H]1CC[C@H](CC1)NC1=NC(=C2N=CN(C2=N1)C1CCCC1)NNCCO JYEDZKFFPWKMOJ-JOCQHMNTSA-N 0.000 description 1
- NKKOFTRGEZKEOF-QAQDUYKDSA-N N[C@H](CC1)CC[C@@H]1NC1=NC(NCC2=CC=NC=C2)=C2N=CN(C3CCCC3)C2=N1 Chemical compound N[C@H](CC1)CC[C@@H]1NC1=NC(NCC2=CC=NC=C2)=C2N=CN(C3CCCC3)C2=N1 NKKOFTRGEZKEOF-QAQDUYKDSA-N 0.000 description 1
- UFPYJZPVCKQWBG-HDJSIYSDSA-N N[C@H]1CC[C@@H](CC1)Nc1nc(NCCCO)c2ncn(C3CCCC3)c2n1 Chemical compound N[C@H]1CC[C@@H](CC1)Nc1nc(NCCCO)c2ncn(C3CCCC3)c2n1 UFPYJZPVCKQWBG-HDJSIYSDSA-N 0.000 description 1
- XBJNACYCCXJNGA-WGSAOQKQSA-N N[C@H]1CC[C@@H](CC1)Nc1nc(NCCc2ccccc2)c2ncn(C3CCCC3)c2n1 Chemical compound N[C@H]1CC[C@@H](CC1)Nc1nc(NCCc2ccccc2)c2ncn(C3CCCC3)c2n1 XBJNACYCCXJNGA-WGSAOQKQSA-N 0.000 description 1
- VENIFOMWOOLMHF-IYARVYRRSA-N N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2ccc(Cl)cc2)c2ncn(C3CCCC3)c2n1 Chemical compound N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2ccc(Cl)cc2)c2ncn(C3CCCC3)c2n1 VENIFOMWOOLMHF-IYARVYRRSA-N 0.000 description 1
- PYVLHYAEYFTCJL-IYARVYRRSA-N N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2ccc(F)cc2)c2ncn(C3CCCC3)c2n1 Chemical compound N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2ccc(F)cc2)c2ncn(C3CCCC3)c2n1 PYVLHYAEYFTCJL-IYARVYRRSA-N 0.000 description 1
- RVGGTBOHOHAWIE-QAQDUYKDSA-N N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2ccccc2F)c2ncn(C3CCCC3)c2n1 Chemical compound N[C@H]1CC[C@@H](CC1)Nc1nc(NCc2ccccc2F)c2ncn(C3CCCC3)c2n1 RVGGTBOHOHAWIE-QAQDUYKDSA-N 0.000 description 1
- WEPPYZKZIHHZIQ-WGSAOQKQSA-N N[C@H]1CC[C@@H](CC1)Nc1nc(NNCCc2ccccc2)c2ncn(C3CCCC3)c2n1 Chemical compound N[C@H]1CC[C@@H](CC1)Nc1nc(NNCCc2ccccc2)c2ncn(C3CCCC3)c2n1 WEPPYZKZIHHZIQ-WGSAOQKQSA-N 0.000 description 1
- 102000048850 Neoplasm Genes Human genes 0.000 description 1
- 108700019961 Neoplasm Genes Proteins 0.000 description 1
- 201000004404 Neurofibroma Diseases 0.000 description 1
- 244000061176 Nicotiana tabacum Species 0.000 description 1
- 108020004485 Nonsense Codon Proteins 0.000 description 1
- 102000043276 Oncogene Human genes 0.000 description 1
- 241000283973 Oryctolagus cuniculus Species 0.000 description 1
- 208000010191 Osteitis Deformans Diseases 0.000 description 1
- 208000000035 Osteochondroma Diseases 0.000 description 1
- 208000001132 Osteoporosis Diseases 0.000 description 1
- 102000004316 Oxidoreductases Human genes 0.000 description 1
- 229910019142 PO4 Inorganic materials 0.000 description 1
- 208000027067 Paget disease of bone Diseases 0.000 description 1
- 208000025618 Paget disease of nipple Diseases 0.000 description 1
- 208000024024 Paget disease of the nipple Diseases 0.000 description 1
- 206010033661 Pancytopenia Diseases 0.000 description 1
- RMUCZJUITONUFY-UHFFFAOYSA-N Phenelzine Chemical compound NNCCC1=CC=CC=C1 RMUCZJUITONUFY-UHFFFAOYSA-N 0.000 description 1
- BHHGXPLMPWCGHP-UHFFFAOYSA-N Phenethylamine Chemical compound NCCC1=CC=CC=C1 BHHGXPLMPWCGHP-UHFFFAOYSA-N 0.000 description 1
- BELBBZDIHDAJOR-UHFFFAOYSA-N Phenolsulfonephthalein Chemical compound C1=CC(O)=CC=C1C1(C=2C=CC(O)=CC=2)C2=CC=CC=C2S(=O)(=O)O1 BELBBZDIHDAJOR-UHFFFAOYSA-N 0.000 description 1
- 108010089430 Phosphoproteins Proteins 0.000 description 1
- 102000007982 Phosphoproteins Human genes 0.000 description 1
- 208000007641 Pinealoma Diseases 0.000 description 1
- 208000007913 Pituitary Neoplasms Diseases 0.000 description 1
- 201000005746 Pituitary adenoma Diseases 0.000 description 1
- 206010061538 Pituitary tumour benign Diseases 0.000 description 1
- 208000020424 Polyglandular disease Diseases 0.000 description 1
- 208000007541 Preleukemia Diseases 0.000 description 1
- 102100025803 Progesterone receptor Human genes 0.000 description 1
- 102100036691 Proliferating cell nuclear antigen Human genes 0.000 description 1
- WUGQZFFCHPXWKQ-UHFFFAOYSA-N Propanolamine Chemical compound NCCCO WUGQZFFCHPXWKQ-UHFFFAOYSA-N 0.000 description 1
- 102000003923 Protein Kinase C Human genes 0.000 description 1
- 108090000315 Protein Kinase C Proteins 0.000 description 1
- 206010056626 Pseudopolyp Diseases 0.000 description 1
- 239000005700 Putrescine Substances 0.000 description 1
- 101000902592 Pyrococcus furiosus (strain ATCC 43587 / DSM 3638 / JCM 8422 / Vc1) DNA polymerase Proteins 0.000 description 1
- 239000012980 RPMI-1640 medium Substances 0.000 description 1
- 102100022151 Ragulator complex protein LAMTOR1 Human genes 0.000 description 1
- 241000773293 Rappaport Species 0.000 description 1
- 208000037323 Rare tumor Diseases 0.000 description 1
- 102000007056 Recombinant Fusion Proteins Human genes 0.000 description 1
- 108010008281 Recombinant Fusion Proteins Proteins 0.000 description 1
- 208000009527 Refractory anemia Diseases 0.000 description 1
- 206010072684 Refractory cytopenia with unilineage dysplasia Diseases 0.000 description 1
- 108050002653 Retinoblastoma protein Proteins 0.000 description 1
- 101150112625 SSN3 gene Proteins 0.000 description 1
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 1
- 101100150415 Schizosaccharomyces pombe (strain 972 / ATCC 24843) srb10 gene Proteins 0.000 description 1
- 101100368917 Schizosaccharomyces pombe (strain 972 / ATCC 24843) taz1 gene Proteins 0.000 description 1
- 208000000453 Skin Neoplasms Diseases 0.000 description 1
- 206010041660 Splenomegaly Diseases 0.000 description 1
- 208000005718 Stomach Neoplasms Diseases 0.000 description 1
- 108010090804 Streptavidin Proteins 0.000 description 1
- KDYFGRWQOYBRFD-UHFFFAOYSA-N Succinic acid Natural products OC(=O)CCC(O)=O KDYFGRWQOYBRFD-UHFFFAOYSA-N 0.000 description 1
- 208000031673 T-Cell Cutaneous Lymphoma Diseases 0.000 description 1
- 208000000389 T-cell leukemia Diseases 0.000 description 1
- 208000027585 T-cell non-Hodgkin lymphoma Diseases 0.000 description 1
- 102000006467 TATA-Box Binding Protein Human genes 0.000 description 1
- 108010044281 TATA-Box Binding Protein Proteins 0.000 description 1
- 206010043276 Teratoma Diseases 0.000 description 1
- 101000652566 Tetrahymena thermophila (strain SB210) Telomerase-associated protein of 19 kDa Proteins 0.000 description 1
- AYFVYJQAPQTCCC-UHFFFAOYSA-N Threonine Natural products CC(O)C(N)C(O)=O AYFVYJQAPQTCCC-UHFFFAOYSA-N 0.000 description 1
- 239000004473 Threonine Substances 0.000 description 1
- 108010022394 Threonine synthase Proteins 0.000 description 1
- 208000005485 Thrombocytosis Diseases 0.000 description 1
- 102000006601 Thymidine Kinase Human genes 0.000 description 1
- 108020004440 Thymidine kinase Proteins 0.000 description 1
- 102000002463 Transcription Factor TFIIIB Human genes 0.000 description 1
- 108010068071 Transcription Factor TFIIIB Proteins 0.000 description 1
- 229920004890 Triton X-100 Polymers 0.000 description 1
- 239000013504 Triton X-100 Substances 0.000 description 1
- 108700025716 Tumor Suppressor Genes Proteins 0.000 description 1
- 102000044209 Tumor Suppressor Genes Human genes 0.000 description 1
- 108010040002 Tumor Suppressor Proteins Proteins 0.000 description 1
- 102000001742 Tumor Suppressor Proteins Human genes 0.000 description 1
- 208000004062 Tumor Virus Infections Diseases 0.000 description 1
- DZGWFCGJZKJUFP-UHFFFAOYSA-N Tyramine Natural products NCCC1=CC=C(O)C=C1 DZGWFCGJZKJUFP-UHFFFAOYSA-N 0.000 description 1
- 208000015778 Undifferentiated pleomorphic sarcoma Diseases 0.000 description 1
- 240000006064 Urena lobata Species 0.000 description 1
- 206010046798 Uterine leiomyoma Diseases 0.000 description 1
- 208000009311 VIPoma Diseases 0.000 description 1
- 102000013127 Vimentin Human genes 0.000 description 1
- 108010065472 Vimentin Proteins 0.000 description 1
- JXLYSJRDGCGARV-WWYNWVTFSA-N Vinblastine Natural products O=C(O[C@H]1[C@](O)(C(=O)OC)[C@@H]2N(C)c3c(cc(c(OC)c3)[C@]3(C(=O)OC)c4[nH]c5c(c4CCN4C[C@](O)(CC)C[C@H](C3)C4)cccc5)[C@@]32[C@H]2[C@@]1(CC)C=CCN2CC3)C JXLYSJRDGCGARV-WWYNWVTFSA-N 0.000 description 1
- 229940122803 Vinca alkaloid Drugs 0.000 description 1
- BZHJMEDXRYGGRV-UHFFFAOYSA-N Vinyl chloride Chemical compound ClC=C BZHJMEDXRYGGRV-UHFFFAOYSA-N 0.000 description 1
- 208000016025 Waldenstroem macroglobulinemia Diseases 0.000 description 1
- 208000008383 Wilms tumor Diseases 0.000 description 1
- 206010048214 Xanthoma Diseases 0.000 description 1
- 206010048215 Xanthomatosis Diseases 0.000 description 1
- 101100459258 Xenopus laevis myc-a gene Proteins 0.000 description 1
- 241000607479 Yersinia pestis Species 0.000 description 1
- 201000008629 Zollinger-Ellison syndrome Diseases 0.000 description 1
- FPVUCBMBMUHRDX-XMMPIXPASA-N [4-hydroxy-3-(3-methylbut-2-enyl)phenyl]methyl (2r)-4-hydroxy-3-(4-hydroxyphenyl)-2-methyl-5-oxofuran-2-carboxylate Chemical compound C1=C(O)C(CC=C(C)C)=CC(COC(=O)[C@@]2(C)C(=C(O)C(=O)O2)C=2C=CC(O)=CC=2)=C1 FPVUCBMBMUHRDX-XMMPIXPASA-N 0.000 description 1
- 230000005856 abnormality Effects 0.000 description 1
- 238000002835 absorbance Methods 0.000 description 1
- 150000007513 acids Chemical class 0.000 description 1
- 230000001154 acute effect Effects 0.000 description 1
- 208000021841 acute erythroid leukemia Diseases 0.000 description 1
- 210000002534 adenoid Anatomy 0.000 description 1
- 210000004100 adrenal gland Anatomy 0.000 description 1
- 125000002723 alicyclic group Chemical group 0.000 description 1
- 229940100198 alkylating agent Drugs 0.000 description 1
- 239000002168 alkylating agent Substances 0.000 description 1
- 235000020224 almond Nutrition 0.000 description 1
- VREFGVBLTWBCJP-UHFFFAOYSA-N alprazolam Chemical compound C12=CC(Cl)=CC=C2N2C(C)=NN=C2CN=C1C1=CC=CC=C1 VREFGVBLTWBCJP-UHFFFAOYSA-N 0.000 description 1
- 230000004075 alteration Effects 0.000 description 1
- 230000031016 anaphase Effects 0.000 description 1
- 239000003098 androgen Substances 0.000 description 1
- 108010080146 androgen receptors Proteins 0.000 description 1
- 229940030486 androgens Drugs 0.000 description 1
- 208000007502 anemia Diseases 0.000 description 1
- 239000003242 anti bacterial agent Substances 0.000 description 1
- 230000000340 anti-metabolite Effects 0.000 description 1
- 230000001028 anti-proliverative effect Effects 0.000 description 1
- 229940088710 antibiotic agent Drugs 0.000 description 1
- 229940100197 antimetabolite Drugs 0.000 description 1
- 239000002256 antimetabolite Substances 0.000 description 1
- 229940045719 antineoplastic alkylating agent nitrosoureas Drugs 0.000 description 1
- 239000000010 aprotic solvent Substances 0.000 description 1
- 229960004405 aprotinin Drugs 0.000 description 1
- 229910052785 arsenic Inorganic materials 0.000 description 1
- RQNWIZPPADIBDY-UHFFFAOYSA-N arsenic atom Chemical compound [As] RQNWIZPPADIBDY-UHFFFAOYSA-N 0.000 description 1
- 230000005744 arteriovenous malformation Effects 0.000 description 1
- 229960001230 asparagine Drugs 0.000 description 1
- 235000009582 asparagine Nutrition 0.000 description 1
- 125000004429 atom Chemical group 0.000 description 1
- QVGXLLKOCUKJST-UHFFFAOYSA-N atomic oxygen Chemical compound [O] QVGXLLKOCUKJST-UHFFFAOYSA-N 0.000 description 1
- 108700042656 bcl-1 Genes Proteins 0.000 description 1
- WPYMKLBDIGXBTP-UHFFFAOYSA-N benzoic acid group Chemical group C(C1=CC=CC=C1)(=O)O WPYMKLBDIGXBTP-UHFFFAOYSA-N 0.000 description 1
- IQFYYKKMVGJFEH-UHFFFAOYSA-N beta-L-thymidine Natural products O=C1NC(=O)C(C)=CN1C1OC(CO)C(O)C1 IQFYYKKMVGJFEH-UHFFFAOYSA-N 0.000 description 1
- 230000001588 bifunctional effect Effects 0.000 description 1
- 210000000013 bile duct Anatomy 0.000 description 1
- 230000031018 biological processes and functions Effects 0.000 description 1
- HRQGCQVOJVTVLU-UHFFFAOYSA-N bis(chloromethyl) ether Chemical class ClCOCCl HRQGCQVOJVTVLU-UHFFFAOYSA-N 0.000 description 1
- 230000000740 bleeding effect Effects 0.000 description 1
- 229960001561 bleomycin Drugs 0.000 description 1
- OYVAGSVQBOHSSS-UAPAGMARSA-O bleomycin A2 Chemical compound N([C@H](C(=O)N[C@H](C)[C@@H](O)[C@H](C)C(=O)N[C@@H]([C@H](O)C)C(=O)NCCC=1SC=C(N=1)C=1SC=C(N=1)C(=O)NCCC[S+](C)C)[C@@H](O[C@H]1[C@H]([C@@H](O)[C@H](O)[C@H](CO)O1)O[C@@H]1[C@H]([C@@H](OC(N)=O)[C@H](O)[C@@H](CO)O1)O)C=1N=CNC=1)C(=O)C1=NC([C@H](CC(N)=O)NC[C@H](N)C(N)=O)=NC(N)=C1C OYVAGSVQBOHSSS-UAPAGMARSA-O 0.000 description 1
- 230000017531 blood circulation Effects 0.000 description 1
- 230000037396 body weight Effects 0.000 description 1
- 238000009835 boiling Methods 0.000 description 1
- 208000016738 bone Paget disease Diseases 0.000 description 1
- 201000007327 bone benign neoplasm Diseases 0.000 description 1
- 229940098773 bovine serum albumin Drugs 0.000 description 1
- QXZGBUJJYSLZLT-FDISYFBBSA-N bradykinin Chemical compound NC(=N)NCCC[C@H](N)C(=O)N1CCC[C@H]1C(=O)N1[C@H](C(=O)NCC(=O)N[C@@H](CC=2C=CC=CC=2)C(=O)N[C@@H](CO)C(=O)N2[C@@H](CCC2)C(=O)N[C@@H](CC=2C=CC=CC=2)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O)CCC1 QXZGBUJJYSLZLT-FDISYFBBSA-N 0.000 description 1
- GDTBXPJZTBHREO-UHFFFAOYSA-N bromine Substances BrBr GDTBXPJZTBHREO-UHFFFAOYSA-N 0.000 description 1
- 229910052794 bromium Inorganic materials 0.000 description 1
- KDYFGRWQOYBRFD-NUQCWPJISA-N butanedioic acid Chemical compound O[14C](=O)CC[14C](O)=O KDYFGRWQOYBRFD-NUQCWPJISA-N 0.000 description 1
- 125000003178 carboxy group Chemical group [H]OC(*)=O 0.000 description 1
- 208000002458 carcinoid tumor Diseases 0.000 description 1
- 229960005243 carmustine Drugs 0.000 description 1
- 230000003197 catalytic effect Effects 0.000 description 1
- 108700021031 cdc Genes Proteins 0.000 description 1
- 230000018486 cell cycle phase Effects 0.000 description 1
- 230000006369 cell cycle progression Effects 0.000 description 1
- 230000003915 cell function Effects 0.000 description 1
- 239000008004 cell lysis buffer Substances 0.000 description 1
- 230000004640 cellular pathway Effects 0.000 description 1
- 229940106157 cellulase Drugs 0.000 description 1
- 201000007455 central nervous system cancer Diseases 0.000 description 1
- 201000004226 ceruminoma Diseases 0.000 description 1
- AOXOCDRNSPFDPE-UKEONUMOSA-N chembl413654 Chemical compound C([C@H](C(=O)NCC(=O)N[C@H](CC=1C2=CC=CC=C2NC=1)C(=O)N[C@H](CCSC)C(=O)N[C@H](CC(O)=O)C(=O)N[C@H](CC=1C=CC=CC=1)C(N)=O)NC(=O)[C@@H](C)NC(=O)[C@@H](CCC(O)=O)NC(=O)[C@@H](CCC(O)=O)NC(=O)[C@@H](CCC(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CC=1C2=CC=CC=C2NC=1)NC(=O)[C@H]1N(CCC1)C(=O)CNC(=O)[C@@H](N)CCC(O)=O)C1=CC=C(O)C=C1 AOXOCDRNSPFDPE-UKEONUMOSA-N 0.000 description 1
- 239000013043 chemical agent Substances 0.000 description 1
- 239000003795 chemical substances by application Substances 0.000 description 1
- PRQROPMIIGLWRP-BZSNNMDCSA-N chemotactic peptide Chemical compound CSCC[C@H](NC=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 PRQROPMIIGLWRP-BZSNNMDCSA-N 0.000 description 1
- 210000000038 chest Anatomy 0.000 description 1
- 229940112822 chewing gum Drugs 0.000 description 1
- 235000015218 chewing gum Nutrition 0.000 description 1
- 235000013330 chicken meat Nutrition 0.000 description 1
- 229910052801 chlorine Inorganic materials 0.000 description 1
- 208000006990 cholangiocarcinoma Diseases 0.000 description 1
- 201000005217 chondroblastoma Diseases 0.000 description 1
- 210000003737 chromaffin cell Anatomy 0.000 description 1
- ZCDOYSPFYFSLEW-UHFFFAOYSA-N chromate(2-) Chemical class [O-][Cr]([O-])(=O)=O ZCDOYSPFYFSLEW-UHFFFAOYSA-N 0.000 description 1
- 230000010428 chromatin condensation Effects 0.000 description 1
- 208000016350 chronic hepatitis B virus infection Diseases 0.000 description 1
- 201000010902 chronic myelomonocytic leukemia Diseases 0.000 description 1
- 235000019504 cigarettes Nutrition 0.000 description 1
- 238000004140 cleaning Methods 0.000 description 1
- 238000003776 cleavage reaction Methods 0.000 description 1
- 238000011260 co-administration Methods 0.000 description 1
- 230000000295 complement effect Effects 0.000 description 1
- 229940124301 concurrent medication Drugs 0.000 description 1
- 239000000470 constituent Substances 0.000 description 1
- 230000001054 cortical effect Effects 0.000 description 1
- 210000003618 cortical neuron Anatomy 0.000 description 1
- 239000012043 crude product Substances 0.000 description 1
- 239000013078 crystal Substances 0.000 description 1
- 201000007241 cutaneous T cell lymphoma Diseases 0.000 description 1
- 208000035250 cutaneous malignant susceptibility to 1 melanoma Diseases 0.000 description 1
- 239000002875 cyclin dependent kinase inhibitor Substances 0.000 description 1
- 125000001995 cyclobutyl group Chemical group [H]C1([H])C([H])([H])C([H])(*)C1([H])[H] 0.000 description 1
- 229960002944 cyclofenil Drugs 0.000 description 1
- 125000000582 cycloheptyl group Chemical group [H]C1([H])C([H])([H])C([H])([H])C([H])([H])C([H])(*)C([H])([H])C1([H])[H] 0.000 description 1
- 125000000113 cyclohexyl group Chemical group [H]C1([H])C([H])([H])C([H])([H])C([H])(*)C([H])([H])C1([H])[H] 0.000 description 1
- AVKNGPAMCBSNSO-UHFFFAOYSA-N cyclohexylmethanamine Chemical compound NCC1CCCCC1 AVKNGPAMCBSNSO-UHFFFAOYSA-N 0.000 description 1
- 125000000640 cyclooctyl group Chemical group [H]C1([H])C([H])([H])C([H])([H])C([H])([H])C([H])(*)C([H])([H])C([H])([H])C1([H])[H] 0.000 description 1
- 229960004397 cyclophosphamide Drugs 0.000 description 1
- 229960000684 cytarabine Drugs 0.000 description 1
- 230000000093 cytochemical effect Effects 0.000 description 1
- 230000021953 cytokinesis Effects 0.000 description 1
- 230000009089 cytolysis Effects 0.000 description 1
- 208000024389 cytopenia Diseases 0.000 description 1
- 210000000805 cytoplasm Anatomy 0.000 description 1
- 230000001086 cytosolic effect Effects 0.000 description 1
- 229940127089 cytotoxic agent Drugs 0.000 description 1
- GYOZYWVXFNDGLU-XLPZGREQSA-N dTMP Chemical compound O=C1NC(=O)C(C)=CN1[C@@H]1O[C@H](COP(O)(O)=O)[C@@H](O)C1 GYOZYWVXFNDGLU-XLPZGREQSA-N 0.000 description 1
- STQGQHZAVUOBTE-VGBVRHCVSA-N daunorubicin Chemical compound O([C@H]1C[C@@](O)(CC=2C(O)=C3C(=O)C=4C=CC=C(C=4C(=O)C3=C(O)C=21)OC)C(C)=O)[C@H]1C[C@H](N)[C@H](O)[C@H](C)O1 STQGQHZAVUOBTE-VGBVRHCVSA-N 0.000 description 1
- 230000007423 decrease Effects 0.000 description 1
- 230000007850 degeneration Effects 0.000 description 1
- 230000003412 degenerative effect Effects 0.000 description 1
- 230000030609 dephosphorylation Effects 0.000 description 1
- 238000006209 dephosphorylation reaction Methods 0.000 description 1
- 230000002074 deregulated effect Effects 0.000 description 1
- 210000005045 desmin Anatomy 0.000 description 1
- 238000001514 detection method Methods 0.000 description 1
- 102000004419 dihydrofolate reductase Human genes 0.000 description 1
- LOKCTEFSRHRXRJ-UHFFFAOYSA-I dipotassium trisodium dihydrogen phosphate hydrogen phosphate dichloride Chemical compound P(=O)(O)(O)[O-].[K+].P(=O)(O)([O-])[O-].[Na+].[Na+].[Cl-].[K+].[Cl-].[Na+] LOKCTEFSRHRXRJ-UHFFFAOYSA-I 0.000 description 1
- BNIILDVGGAEEIG-UHFFFAOYSA-L disodium hydrogen phosphate Chemical compound [Na+].[Na+].OP([O-])([O-])=O BNIILDVGGAEEIG-UHFFFAOYSA-L 0.000 description 1
- 238000010494 dissociation reaction Methods 0.000 description 1
- 230000005593 dissociations Effects 0.000 description 1
- 210000004921 distal colon Anatomy 0.000 description 1
- VHJLVAABSRFDPM-QWWZWVQMSA-N dithiothreitol Chemical compound SC[C@@H](O)[C@H](O)CS VHJLVAABSRFDPM-QWWZWVQMSA-N 0.000 description 1
- 230000003828 downregulation Effects 0.000 description 1
- 229960004679 doxorubicin Drugs 0.000 description 1
- 238000001035 drying Methods 0.000 description 1
- 230000002183 duodenal effect Effects 0.000 description 1
- 210000000613 ear canal Anatomy 0.000 description 1
- 210000000883 ear external Anatomy 0.000 description 1
- 210000000959 ear middle Anatomy 0.000 description 1
- 201000003511 ectopic pregnancy Diseases 0.000 description 1
- 230000002500 effect on skin Effects 0.000 description 1
- 238000010828 elution Methods 0.000 description 1
- 238000005538 encapsulation Methods 0.000 description 1
- 210000003372 endocrine gland Anatomy 0.000 description 1
- 201000011523 endocrine gland cancer Diseases 0.000 description 1
- 238000009261 endocrine therapy Methods 0.000 description 1
- 229940034984 endocrine therapy antineoplastic and immunomodulating agent Drugs 0.000 description 1
- 210000003059 ependyma Anatomy 0.000 description 1
- 230000001667 episodic effect Effects 0.000 description 1
- XWBDWHCCBGMXKG-UHFFFAOYSA-N ethanamine;hydron;chloride Chemical compound Cl.CCN XWBDWHCCBGMXKG-UHFFFAOYSA-N 0.000 description 1
- 210000003527 eukaryotic cell Anatomy 0.000 description 1
- 230000005284 excitation Effects 0.000 description 1
- 201000001343 fallopian tube carcinoma Diseases 0.000 description 1
- 230000008713 feedback mechanism Effects 0.000 description 1
- 230000001605 fetal effect Effects 0.000 description 1
- 210000002950 fibroblast Anatomy 0.000 description 1
- 239000011737 fluorine Substances 0.000 description 1
- 229910052731 fluorine Inorganic materials 0.000 description 1
- 229960002949 fluorouracil Drugs 0.000 description 1
- 238000011010 flushing procedure Methods 0.000 description 1
- 239000004052 folic acid antagonist Substances 0.000 description 1
- 230000003325 follicular Effects 0.000 description 1
- 238000009472 formulation Methods 0.000 description 1
- 208000024386 fungal infectious disease Diseases 0.000 description 1
- DDRPCXLAQZKBJP-UHFFFAOYSA-N furfurylamine Chemical compound NCC1=CC=CO1 DDRPCXLAQZKBJP-UHFFFAOYSA-N 0.000 description 1
- 108020001507 fusion proteins Proteins 0.000 description 1
- 102000037865 fusion proteins Human genes 0.000 description 1
- 208000010749 gastric carcinoma Diseases 0.000 description 1
- 230000002496 gastric effect Effects 0.000 description 1
- 201000000052 gastrinoma Diseases 0.000 description 1
- 238000012252 genetic analysis Methods 0.000 description 1
- 210000004907 gland Anatomy 0.000 description 1
- 229960003180 glutathione Drugs 0.000 description 1
- 239000003102 growth factor Substances 0.000 description 1
- 230000007773 growth pattern Effects 0.000 description 1
- 150000002367 halogens Chemical group 0.000 description 1
- 208000018578 heart valve disease Diseases 0.000 description 1
- 201000002222 hemangioblastoma Diseases 0.000 description 1
- 201000001505 hemoglobinuria Diseases 0.000 description 1
- 208000002672 hepatitis B Diseases 0.000 description 1
- 208000006359 hepatoblastoma Diseases 0.000 description 1
- 201000002735 hepatocellular adenoma Diseases 0.000 description 1
- 210000003494 hepatocyte Anatomy 0.000 description 1
- 125000003187 heptyl group Chemical group [H]C([*])([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])[H] 0.000 description 1
- 230000002363 herbicidal effect Effects 0.000 description 1
- 239000004009 herbicide Substances 0.000 description 1
- 239000000833 heterodimer Substances 0.000 description 1
- 125000004051 hexyl group Chemical group [H]C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])* 0.000 description 1
- 229960001340 histamine Drugs 0.000 description 1
- 102000047723 human CCND1 Human genes 0.000 description 1
- PYFDZOCGFHIRST-UHFFFAOYSA-N hydron;(3-iodophenyl)methanamine;chloride Chemical compound Cl.NCC1=CC=CC(I)=C1 PYFDZOCGFHIRST-UHFFFAOYSA-N 0.000 description 1
- ZTUJDPKOHPKRMO-UHFFFAOYSA-N hydron;2,2,2-trifluoroethanamine;chloride Chemical compound Cl.NCC(F)(F)F ZTUJDPKOHPKRMO-UHFFFAOYSA-N 0.000 description 1
- 230000006951 hyperphosphorylation Effects 0.000 description 1
- 206010020718 hyperplasia Diseases 0.000 description 1
- 208000017819 hyperplastic polyp Diseases 0.000 description 1
- 201000002312 ileal neoplasm Diseases 0.000 description 1
- 210000003405 ileum Anatomy 0.000 description 1
- 102000018358 immunoglobulin Human genes 0.000 description 1
- 229960003444 immunosuppressant agent Drugs 0.000 description 1
- 239000003018 immunosuppressive agent Substances 0.000 description 1
- 238000000099 in vitro assay Methods 0.000 description 1
- 238000005462 in vivo assay Methods 0.000 description 1
- 238000011065 in-situ storage Methods 0.000 description 1
- 238000011534 incubation Methods 0.000 description 1
- 230000002458 infectious effect Effects 0.000 description 1
- 230000008595 infiltration Effects 0.000 description 1
- 238000001764 infiltration Methods 0.000 description 1
- ZPNFWUPYTFPOJU-LPYSRVMUSA-N iniprol Chemical compound C([C@H]1C(=O)NCC(=O)NCC(=O)N[C@H]2CSSC[C@H]3C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](C)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@H](C(N[C@H](C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC=4C=CC(O)=CC=4)C(=O)N[C@@H](CC=4C=CC=CC=4)C(=O)N[C@@H](CC=4C=CC(O)=CC=4)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](C)C(=O)NCC(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CSSC[C@H](NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](C)NC(=O)[C@H](CO)NC(=O)[C@H](CCCCN)NC(=O)[C@H](CC=4C=CC=CC=4)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@H](CCCCN)NC(=O)[C@H](C)NC(=O)[C@H](CCCNC(N)=N)NC2=O)C(=O)N[C@@H](CCSC)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CSSC[C@H](NC(=O)[C@H](CC=2C=CC=CC=2)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H]2N(CCC2)C(=O)[C@@H](N)CCCNC(N)=N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(O)=O)C(=O)N2[C@@H](CCC2)C(=O)N2[C@@H](CCC2)C(=O)N[C@@H](CC=2C=CC(O)=CC=2)C(=O)N[C@@H]([C@@H](C)O)C(=O)NCC(=O)N2[C@@H](CCC2)C(=O)N3)C(=O)NCC(=O)NCC(=O)N[C@@H](C)C(O)=O)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@H](C(=O)N[C@@H](CC=2C=CC=CC=2)C(=O)N[C@H](C(=O)N1)C(C)C)[C@@H](C)O)[C@@H](C)CC)=O)[C@@H](C)CC)C1=CC=C(O)C=C1 ZPNFWUPYTFPOJU-LPYSRVMUSA-N 0.000 description 1
- 229910001410 inorganic ion Inorganic materials 0.000 description 1
- 238000003780 insertion Methods 0.000 description 1
- 239000002198 insoluble material Substances 0.000 description 1
- 230000003993 interaction Effects 0.000 description 1
- 210000003963 intermediate filament Anatomy 0.000 description 1
- 230000016507 interphase Effects 0.000 description 1
- 230000000968 intestinal effect Effects 0.000 description 1
- 210000000936 intestine Anatomy 0.000 description 1
- 210000003228 intrahepatic bile duct Anatomy 0.000 description 1
- 238000007918 intramuscular administration Methods 0.000 description 1
- 238000001990 intravenous administration Methods 0.000 description 1
- 230000005865 ionizing radiation Effects 0.000 description 1
- 208000037906 ischaemic injury Diseases 0.000 description 1
- 210000004153 islets of langerhan Anatomy 0.000 description 1
- 238000002955 isolation Methods 0.000 description 1
- 125000001972 isopentyl group Chemical group [H]C([H])([H])C([H])(C([H])([H])[H])C([H])([H])C([H])([H])* 0.000 description 1
- BMFVGAAISNGQNM-UHFFFAOYSA-N isopentylamine Chemical compound CC(C)CCN BMFVGAAISNGQNM-UHFFFAOYSA-N 0.000 description 1
- 201000009592 jejunal neoplasm Diseases 0.000 description 1
- 210000001630 jejunum Anatomy 0.000 description 1
- 208000020123 juvenile polyp Diseases 0.000 description 1
- 238000011005 laboratory method Methods 0.000 description 1
- 239000004310 lactic acid Substances 0.000 description 1
- 235000014655 lactic acid Nutrition 0.000 description 1
- 208000003849 large cell carcinoma Diseases 0.000 description 1
- 210000000867 larynx Anatomy 0.000 description 1
- 230000003902 lesion Effects 0.000 description 1
- 210000000265 leukocyte Anatomy 0.000 description 1
- GDBQQVLCIARPGH-ULQDDVLXSA-N leupeptin Chemical compound CC(C)C[C@H](NC(C)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@H](C=O)CCCN=C(N)N GDBQQVLCIARPGH-ULQDDVLXSA-N 0.000 description 1
- 108010052968 leupeptin Proteins 0.000 description 1
- 208000012987 lip and oral cavity carcinoma Diseases 0.000 description 1
- 150000002632 lipids Chemical class 0.000 description 1
- 201000004601 liver benign neoplasm Diseases 0.000 description 1
- 201000011059 lobular neoplasia Diseases 0.000 description 1
- 230000004807 localization Effects 0.000 description 1
- 229960002247 lomustine Drugs 0.000 description 1
- 230000005923 long-lasting effect Effects 0.000 description 1
- 230000001926 lymphatic effect Effects 0.000 description 1
- 210000004324 lymphatic system Anatomy 0.000 description 1
- 230000000527 lymphocytic effect Effects 0.000 description 1
- 210000005210 lymphoid organ Anatomy 0.000 description 1
- 230000001589 lymphoproliferative effect Effects 0.000 description 1
- 208000025036 lymphosarcoma Diseases 0.000 description 1
- 229910001629 magnesium chloride Inorganic materials 0.000 description 1
- 230000036210 malignancy Effects 0.000 description 1
- 201000002742 malignant choroid melanoma Diseases 0.000 description 1
- 201000004593 malignant giant cell tumor Diseases 0.000 description 1
- 208000026037 malignant tumor of neck Diseases 0.000 description 1
- 210000004962 mammalian cell Anatomy 0.000 description 1
- 208000027202 mammary Paget disease Diseases 0.000 description 1
- 239000003550 marker Substances 0.000 description 1
- 230000010534 mechanism of action Effects 0.000 description 1
- 229960004961 mechlorethamine Drugs 0.000 description 1
- HAWPXGHAZFHHAD-UHFFFAOYSA-N mechlorethamine Chemical compound ClCCN(C)CCCl HAWPXGHAZFHHAD-UHFFFAOYSA-N 0.000 description 1
- 230000009245 menopause Effects 0.000 description 1
- 229960001428 mercaptopurine Drugs 0.000 description 1
- QSHDDOUJBYECFT-UHFFFAOYSA-N mercury Chemical compound [Hg] QSHDDOUJBYECFT-UHFFFAOYSA-N 0.000 description 1
- 229910052753 mercury Inorganic materials 0.000 description 1
- 201000008806 mesenchymal cell neoplasm Diseases 0.000 description 1
- 230000037353 metabolic pathway Effects 0.000 description 1
- 229910052751 metal Inorganic materials 0.000 description 1
- 239000002184 metal Substances 0.000 description 1
- 230000001394 metastastic effect Effects 0.000 description 1
- 206010061289 metastatic neoplasm Diseases 0.000 description 1
- 229940098779 methanesulfonic acid Drugs 0.000 description 1
- 229960000485 methotrexate Drugs 0.000 description 1
- NQMRYBIKMRVZLB-UHFFFAOYSA-N methylamine hydrochloride Chemical compound [Cl-].[NH3+]C NQMRYBIKMRVZLB-UHFFFAOYSA-N 0.000 description 1
- RRIRDPSOCUCGBV-UHFFFAOYSA-N methylenedioxyphenethylamine Chemical compound NCCC1=CC=C2OCOC2=C1 RRIRDPSOCUCGBV-UHFFFAOYSA-N 0.000 description 1
- 239000007758 minimum essential medium Substances 0.000 description 1
- 229960004857 mitomycin Drugs 0.000 description 1
- 230000009456 molecular mechanism Effects 0.000 description 1
- 239000003068 molecular probe Substances 0.000 description 1
- 208000008588 molluscum contagiosum Diseases 0.000 description 1
- 150000004682 monohydrates Chemical class 0.000 description 1
- 230000000877 morphologic effect Effects 0.000 description 1
- 210000004400 mucous membrane Anatomy 0.000 description 1
- 201000003428 multiple mucosal neuroma Diseases 0.000 description 1
- 230000003387 muscular Effects 0.000 description 1
- 201000000050 myeloid neoplasm Diseases 0.000 description 1
- CXGFWBPQQXZELI-UHFFFAOYSA-N n-ethylpyridin-2-amine Chemical compound CCNC1=CC=CC=N1 CXGFWBPQQXZELI-UHFFFAOYSA-N 0.000 description 1
- JNHZPXFARRJZQR-UHFFFAOYSA-N n-ethylpyridin-3-amine Chemical compound CCNC1=CC=CN=C1 JNHZPXFARRJZQR-UHFFFAOYSA-N 0.000 description 1
- OTIDWUALRXXVIM-UHFFFAOYSA-N n-ethylpyridin-4-amine Chemical compound CCNC1=CC=NC=C1 OTIDWUALRXXVIM-UHFFFAOYSA-N 0.000 description 1
- ALBBQFWHUCTZFM-UHFFFAOYSA-N n-methyl-1h-benzimidazol-2-amine Chemical compound C1=CC=C2NC(NC)=NC2=C1 ALBBQFWHUCTZFM-UHFFFAOYSA-N 0.000 description 1
- 210000004897 n-terminal region Anatomy 0.000 description 1
- 210000001989 nasopharynx Anatomy 0.000 description 1
- 125000001971 neopentyl group Chemical group [H]C([*])([H])C(C([H])([H])[H])(C([H])([H])[H])C([H])([H])[H] 0.000 description 1
- 210000005036 nerve Anatomy 0.000 description 1
- 210000000653 nervous system Anatomy 0.000 description 1
- 208000007538 neurilemmoma Diseases 0.000 description 1
- 230000004770 neurodegeneration Effects 0.000 description 1
- 230000000926 neurological effect Effects 0.000 description 1
- 230000019581 neuron apoptotic process Effects 0.000 description 1
- 230000016273 neuron death Effects 0.000 description 1
- 230000006576 neuronal survival Effects 0.000 description 1
- 229910052759 nickel Inorganic materials 0.000 description 1
- 239000012299 nitrogen atmosphere Substances 0.000 description 1
- 230000037434 nonsense mutation Effects 0.000 description 1
- 231100000252 nontoxic Toxicity 0.000 description 1
- 230000003000 nontoxic effect Effects 0.000 description 1
- 210000001331 nose Anatomy 0.000 description 1
- 235000015097 nutrients Nutrition 0.000 description 1
- 230000035764 nutrition Effects 0.000 description 1
- 235000016709 nutrition Nutrition 0.000 description 1
- IOQPZZOEVPZRBK-UHFFFAOYSA-N octan-1-amine Chemical compound CCCCCCCCN IOQPZZOEVPZRBK-UHFFFAOYSA-N 0.000 description 1
- 125000002347 octyl group Chemical group [H]C([*])([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])[H] 0.000 description 1
- 231100000590 oncogenic Toxicity 0.000 description 1
- 230000002246 oncogenic effect Effects 0.000 description 1
- 230000004650 oncogenic pathway Effects 0.000 description 1
- 238000011275 oncology therapy Methods 0.000 description 1
- 210000001328 optic nerve Anatomy 0.000 description 1
- 230000008520 organization Effects 0.000 description 1
- 230000001599 osteoclastic effect Effects 0.000 description 1
- 208000003388 osteoid osteoma Diseases 0.000 description 1
- 230000000010 osteolytic effect Effects 0.000 description 1
- 208000008798 osteoma Diseases 0.000 description 1
- 210000004681 ovum Anatomy 0.000 description 1
- NYGCBQKDTGBHSC-UHFFFAOYSA-N oxan-2-ylmethanamine Chemical compound NCC1CCCCO1 NYGCBQKDTGBHSC-UHFFFAOYSA-N 0.000 description 1
- 239000001301 oxygen Substances 0.000 description 1
- 229910052760 oxygen Inorganic materials 0.000 description 1
- 229940094443 oxytocics prostaglandins Drugs 0.000 description 1
- 108700042657 p16 Genes Proteins 0.000 description 1
- 101800002723 p18 Proteins 0.000 description 1
- 210000000496 pancreas Anatomy 0.000 description 1
- 201000002528 pancreatic cancer Diseases 0.000 description 1
- 208000022102 pancreatic neuroendocrine neoplasm Diseases 0.000 description 1
- 210000003695 paranasal sinus Anatomy 0.000 description 1
- 230000000849 parathyroid Effects 0.000 description 1
- 210000002990 parathyroid gland Anatomy 0.000 description 1
- 230000001314 paroxysmal effect Effects 0.000 description 1
- 230000007170 pathology Effects 0.000 description 1
- 230000000149 penetrating effect Effects 0.000 description 1
- 210000003899 penis Anatomy 0.000 description 1
- 125000001147 pentyl group Chemical group C(CCCC)* 0.000 description 1
- 229940100684 pentylamine Drugs 0.000 description 1
- 208000027232 peripheral nervous system disease Diseases 0.000 description 1
- 208000033808 peripheral neuropathy Diseases 0.000 description 1
- 229940083251 peripheral vasodilators purine derivative Drugs 0.000 description 1
- 229960000964 phenelzine Drugs 0.000 description 1
- 229960003531 phenolsulfonphthalein Drugs 0.000 description 1
- WLJVXDMOQOGPHL-UHFFFAOYSA-N phenylacetic acid Chemical compound OC(=O)CC1=CC=CC=C1 WLJVXDMOQOGPHL-UHFFFAOYSA-N 0.000 description 1
- 239000010452 phosphate Substances 0.000 description 1
- 239000002953 phosphate buffered saline Substances 0.000 description 1
- 230000035790 physiological processes and functions Effects 0.000 description 1
- 208000024724 pineal body neoplasm Diseases 0.000 description 1
- 201000004123 pineal gland cancer Diseases 0.000 description 1
- 208000021310 pituitary gland adenoma Diseases 0.000 description 1
- 230000008635 plant growth Effects 0.000 description 1
- 208000010626 plasma cell neoplasm Diseases 0.000 description 1
- 239000013612 plasmid Substances 0.000 description 1
- 210000004180 plasmocyte Anatomy 0.000 description 1
- 210000001778 pluripotent stem cell Anatomy 0.000 description 1
- 239000002798 polar solvent Substances 0.000 description 1
- 230000008488 polyadenylation Effects 0.000 description 1
- 229920000642 polymer Polymers 0.000 description 1
- 208000014081 polyp of colon Diseases 0.000 description 1
- 210000002729 polyribosome Anatomy 0.000 description 1
- CHKVPAROMQMJNQ-UHFFFAOYSA-M potassium bisulfate Chemical compound [K+].OS([O-])(=O)=O CHKVPAROMQMJNQ-UHFFFAOYSA-M 0.000 description 1
- 229910000343 potassium bisulfate Inorganic materials 0.000 description 1
- 230000003389 potentiating effect Effects 0.000 description 1
- 239000002244 precipitate Substances 0.000 description 1
- 239000002243 precursor Substances 0.000 description 1
- 230000035935 pregnancy Effects 0.000 description 1
- 230000002265 prevention Effects 0.000 description 1
- 208000025638 primary cutaneous T-cell non-Hodgkin lymphoma Diseases 0.000 description 1
- 201000006870 primary polycythemia Diseases 0.000 description 1
- 239000000047 product Substances 0.000 description 1
- 108090000468 progesterone receptors Proteins 0.000 description 1
- 230000000069 prophylactic effect Effects 0.000 description 1
- 238000011321 prophylaxis Methods 0.000 description 1
- 125000004805 propylene group Chemical group [H]C([H])([H])C([H])([*:1])C([H])([H])[*:2] 0.000 description 1
- 150000003180 prostaglandins Chemical class 0.000 description 1
- 201000001514 prostate carcinoma Diseases 0.000 description 1
- 238000000746 purification Methods 0.000 description 1
- 230000006825 purine synthesis Effects 0.000 description 1
- 210000000449 purkinje cell Anatomy 0.000 description 1
- TXQWFIVRZNOPCK-UHFFFAOYSA-N pyridin-4-ylmethanamine Chemical compound NCC1=CC=NC=C1 TXQWFIVRZNOPCK-UHFFFAOYSA-N 0.000 description 1
- 230000006824 pyrimidine synthesis Effects 0.000 description 1
- 229940107700 pyruvic acid Drugs 0.000 description 1
- 238000001959 radiotherapy Methods 0.000 description 1
- 239000011541 reaction mixture Substances 0.000 description 1
- 230000008707 rearrangement Effects 0.000 description 1
- 206010038038 rectal cancer Diseases 0.000 description 1
- 201000001275 rectum cancer Diseases 0.000 description 1
- 230000002787 reinforcement Effects 0.000 description 1
- 238000007634 remodeling Methods 0.000 description 1
- 230000010076 replication Effects 0.000 description 1
- 210000002345 respiratory system Anatomy 0.000 description 1
- 230000004044 response Effects 0.000 description 1
- 230000000717 retained effect Effects 0.000 description 1
- 230000003307 reticuloendothelial effect Effects 0.000 description 1
- 208000029922 reticulum cell sarcoma Diseases 0.000 description 1
- 239000000523 sample Substances 0.000 description 1
- 230000007017 scission Effects 0.000 description 1
- 238000004062 sedimentation Methods 0.000 description 1
- 230000035945 sensitivity Effects 0.000 description 1
- 229940076279 serotonin Drugs 0.000 description 1
- 230000001568 sexual effect Effects 0.000 description 1
- 210000001599 sigmoid colon Anatomy 0.000 description 1
- 230000011664 signaling Effects 0.000 description 1
- 238000010898 silica gel chromatography Methods 0.000 description 1
- 208000014964 small bowel Crohn disease Diseases 0.000 description 1
- 208000000649 small cell carcinoma Diseases 0.000 description 1
- 208000000587 small cell lung carcinoma Diseases 0.000 description 1
- 201000002314 small intestine cancer Diseases 0.000 description 1
- 208000014680 small intestine neoplasm Diseases 0.000 description 1
- 239000011734 sodium Substances 0.000 description 1
- 238000002415 sodium dodecyl sulfate polyacrylamide gel electrophoresis Methods 0.000 description 1
- 210000000952 spleen Anatomy 0.000 description 1
- 206010041823 squamous cell carcinoma Diseases 0.000 description 1
- 239000007858 starting material Substances 0.000 description 1
- 201000011549 stomach cancer Diseases 0.000 description 1
- 201000000498 stomach carcinoma Diseases 0.000 description 1
- 238000007920 subcutaneous administration Methods 0.000 description 1
- 150000003460 sulfonic acids Chemical class 0.000 description 1
- 231100000338 sulforhodamine B assay Toxicity 0.000 description 1
- 230000036561 sun exposure Effects 0.000 description 1
- 239000006228 supernatant Substances 0.000 description 1
- 230000004083 survival effect Effects 0.000 description 1
- 239000008399 tap water Substances 0.000 description 1
- 235000020679 tap water Nutrition 0.000 description 1
- 210000003582 temporal bone Anatomy 0.000 description 1
- 238000012360 testing method Methods 0.000 description 1
- YLQBMQCUIZJEEH-UHFFFAOYSA-N tetrahydrofuran Natural products C=1C=COC=1 YLQBMQCUIZJEEH-UHFFFAOYSA-N 0.000 description 1
- 238000010257 thawing Methods 0.000 description 1
- FKKJJPMGAWGYPN-UHFFFAOYSA-N thiophen-2-ylmethanamine Chemical compound NCC1=CC=CS1 FKKJJPMGAWGYPN-UHFFFAOYSA-N 0.000 description 1
- 125000000341 threoninyl group Chemical group [H]OC([H])(C([H])([H])[H])C([H])(N([H])[H])C(*)=O 0.000 description 1
- 206010043554 thrombocytopenia Diseases 0.000 description 1
- 229940104230 thymidine Drugs 0.000 description 1
- 235000019505 tobacco product Nutrition 0.000 description 1
- 239000002341 toxic gas Substances 0.000 description 1
- 231100000419 toxicity Toxicity 0.000 description 1
- 230000001988 toxicity Effects 0.000 description 1
- 230000001052 transient effect Effects 0.000 description 1
- 230000017105 transposition Effects 0.000 description 1
- 150000003628 tricarboxylic acids Chemical class 0.000 description 1
- YNJBWRMUSHSURL-UHFFFAOYSA-N trichloroacetic acid Chemical compound OC(=O)C(Cl)(Cl)Cl YNJBWRMUSHSURL-UHFFFAOYSA-N 0.000 description 1
- ITMCEJHCFYSIIV-UHFFFAOYSA-N triflic acid Chemical compound OS(=O)(=O)C(F)(F)F ITMCEJHCFYSIIV-UHFFFAOYSA-N 0.000 description 1
- 208000022271 tubular adenoma Diseases 0.000 description 1
- 230000010024 tubular injury Effects 0.000 description 1
- 208000037978 tubular injury Diseases 0.000 description 1
- 208000022158 tubulovillous adenoma Diseases 0.000 description 1
- 230000005740 tumor formation Effects 0.000 description 1
- 229960003732 tyramine Drugs 0.000 description 1
- 125000001493 tyrosinyl group Chemical group [H]OC1=C([H])C([H])=C(C([H])=C1[H])C([H])([H])C([H])(N([H])[H])C(*)=O 0.000 description 1
- 230000004222 uncontrolled growth Effects 0.000 description 1
- 241001430294 unidentified retrovirus Species 0.000 description 1
- 210000000626 ureter Anatomy 0.000 description 1
- 210000003708 urethra Anatomy 0.000 description 1
- 210000001635 urinary tract Anatomy 0.000 description 1
- 210000002700 urine Anatomy 0.000 description 1
- 230000002227 vasoactive effect Effects 0.000 description 1
- 235000013311 vegetables Nutrition 0.000 description 1
- 239000003981 vehicle Substances 0.000 description 1
- 208000009540 villous adenoma Diseases 0.000 description 1
- 210000005048 vimentin Anatomy 0.000 description 1
- 229960003048 vinblastine Drugs 0.000 description 1
- JXLYSJRDGCGARV-XQKSVPLYSA-N vincaleukoblastine Chemical compound C([C@@H](C[C@]1(C(=O)OC)C=2C(=CC3=C([C@]45[C@H]([C@@]([C@H](OC(C)=O)[C@]6(CC)C=CCN([C@H]56)CC4)(O)C(=O)OC)N3C)C=2)OC)C[C@@](C2)(O)CC)N2CCC2=C1NC1=CC=CC=C21 JXLYSJRDGCGARV-XQKSVPLYSA-N 0.000 description 1
- OGWKCGZFUXNPDA-XQKSVPLYSA-N vincristine Chemical compound C([N@]1C[C@@H](C[C@]2(C(=O)OC)C=3C(=CC4=C([C@]56[C@H]([C@@]([C@H](OC(C)=O)[C@]7(CC)C=CCN([C@H]67)CC5)(O)C(=O)OC)N4C=O)C=3)OC)C[C@@](C1)(O)CC)CC1=C2NC2=CC=CC=C12 OGWKCGZFUXNPDA-XQKSVPLYSA-N 0.000 description 1
- 229960004528 vincristine Drugs 0.000 description 1
- OGWKCGZFUXNPDA-UHFFFAOYSA-N vincristine Natural products C1C(CC)(O)CC(CC2(C(=O)OC)C=3C(=CC4=C(C56C(C(C(OC(C)=O)C7(CC)C=CCN(C67)CC5)(O)C(=O)OC)N4C=O)C=3)OC)CN1CCC1=C2NC2=CC=CC=C12 OGWKCGZFUXNPDA-UHFFFAOYSA-N 0.000 description 1
- 210000001835 viscera Anatomy 0.000 description 1
- 208000013013 vulvar carcinoma Diseases 0.000 description 1
- 235000012431 wafers Nutrition 0.000 description 1
- 238000005406 washing Methods 0.000 description 1
Classifications
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07D—HETEROCYCLIC COMPOUNDS
- C07D473/00—Heterocyclic compounds containing purine ring systems
- C07D473/02—Heterocyclic compounds containing purine ring systems with oxygen, sulphur, or nitrogen atoms directly attached in positions 2 and 6
- C07D473/16—Heterocyclic compounds containing purine ring systems with oxygen, sulphur, or nitrogen atoms directly attached in positions 2 and 6 two nitrogen atoms
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P25/00—Drugs for disorders of the nervous system
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P25/00—Drugs for disorders of the nervous system
- A61P25/18—Antipsychotics, i.e. neuroleptics; Drugs for mania or schizophrenia
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
- A61P35/02—Antineoplastic agents specific for leukemia
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P43/00—Drugs for specific purposes, not provided for in groups A61P1/00-A61P41/00
Landscapes
- Health & Medical Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Organic Chemistry (AREA)
- Life Sciences & Earth Sciences (AREA)
- Public Health (AREA)
- General Chemical & Material Sciences (AREA)
- Medicinal Chemistry (AREA)
- Nuclear Medicine, Radiotherapy & Molecular Imaging (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Pharmacology & Pharmacy (AREA)
- Veterinary Medicine (AREA)
- Animal Behavior & Ethology (AREA)
- General Health & Medical Sciences (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Engineering & Computer Science (AREA)
- Biomedical Technology (AREA)
- Neurology (AREA)
- Neurosurgery (AREA)
- Hematology (AREA)
- Oncology (AREA)
- Psychiatry (AREA)
- Pharmaceuticals Containing Other Organic And Inorganic Compounds (AREA)
Description
- Die vorliegende Erfindung betrifft 6,9-disubstituierte 2-[trans-(4-Aminocyclohexyl)amino]purine und Verfahren, die dieselben als antineoplastische Mittel oder zur Behandlung einer neuronalen Verletzung und Degeneration verwenden.
- Hintergrund der Erfindung
- Die Zellteilung, sowohl in normalen als auch neoplastischen Zellen, ist ein streng kontrolliertes Ereignis, das in definierten Stadien abläuft. Ruhende Zellen, die sich nicht aktiv teilen, befinden sich in der G0-Phase, da sie entweder vollständig differenziert sind oder sich im Stadium eines zeitweiligen Ruhezustands befinden. Die erste Phase ist die erste Gap-(G1)-Phase, in der sich die Zellen auf die Synthese von DNA vorbereiten. In der späten G1-Phase, in der es einen Restriktionspunkt oder R-Punkt gibt, erfolgt der Eintritt der Zellen in die S-Phase, während der die DNA-Synthese stattfindet. Nach Abschluss der S-Phase treten die Zellen in die zweite Gap-(G2)-Phase ein, während der die Zellen sich auf die Teilung vorbereiten, welcher die Mitose oder M-Phase folgt.
- Erste Versuche bezüglich der Regulation des Zellzyklus offenbarten die Existenz eines Proteins, das als „Maturation Promoting Factor" (MPF) bezeichnet wird, einem Heterodimer mit Kinaseaktivität. Später offenbarte der Vergleich der nachfolgend identifizierten Proteine und der ihnen zugrunde liegenden Gene eine Familie von Hefe-Genen, die als Kontroll-Gene der Zellteilung (cdc) bekannt sind. Weitere Versuche veranschaulichten, dass einige der cdc-Gene Kinasen kodieren, und sie wurden später als Cyclin-abhängige Kinasen (cdks) bezeichnet. Im Ergebnis dieser Umklassifizierung besitzen einige Zellzyk lus-Proteine Doppelbezeichnungen, wie cdk1, die gleichfalls als cdc2 bekannt ist. Der Kinase-Bestandteil von MPF ist jetzt als p34cdc2 identifiziert worden und die regulatorische Subeinheit von MPF wird jetzt als Cyclin B bezeichnet. Cycline wurden zunächst als Proteine identifiziert, deren Spiegel während des Zellzyklus schwankten und die spezifisch in der Mitosephase abgebaut wurden. Derzeit sind die Cycline A–I und die cdks 1–8 bei Tieren identifiziert worden. Es sind Subtypen von Cyclinen und cdks, wie die Cycline B1 und B2, identifiziert worden, wodurch die Nomenklatur noch komplizierter wird.
- Die nachfolgende Forschung auf dem Gebiet der Zellregulation hat gezeigt, dass die Stadien der Zellteilung teilweise durch die Modulation von Cyclinen und Cyclin-abhängigen Kinasen (cdks) erreicht werden. Cycline regulieren nachfolgend cdks und sie sind durch eine homologe 100 Aminosäureregion charakterisiert, die als „Cyclin-Box" bezeichnet wird, die an der Bindung eines Proteinkinase-Partners beteiligt ist. Cdks sind bezüglich der Sequenz und der Größe (35–40 kDa) eng miteinander verwandt und sie werden als Proteinkinasen definiert, die durch Bindung der Cyclin-regulatorischen Subeinheiten aktiviert werden. Cdks enthalten eine konservierte aktive Spaltstelle von annähernd 300 Aminosäuren, die für alle eukaryontische Proteinkinasen charakteristisch ist. Somit scheinen sowohl die Cycline als auch die cdks hoch konservierte Proteinfamilien zu sein.
- Die Isolierung individueller Cycline und cdks hat des weiteren die Identifizierung der Rollen und Wechselwirkungen jedes Bestandteils in den Zellzyklus-Phasenübergängen ermöglicht. Während des Zellzyklus liegen überschüssige Spiegel an cdks vor. Die Aktivierung von cdks findet nach der Cyclinsynthese und der Bindung an die katalytische cdk-Subeinheit statt, deren Ergebnis die Stimulierung der cdk-Serin/Threonin-Kinaseaktivität ist. Die vollständige cdk-Aktivierung erfordert die Phosphorylierung an einem konservierten Threoninrest, der sich in dem T-Loop befindet, durch eine Cyclin-abhängige Kinase aktivierende Kinase (CAK), die selbst ein cdk/Cyclin-Komplex ist, der aus Cyclin H und cdk7 zusammengesetzt ist, und einem dritten Protein von etwa 32 kDa.
- Die Inaktivierung des cdk-Cyclin-Komplexes kann von der Phosphorylierung eines Threonin- und/oder Tyrosinrests an der ATP-Bindungsstelle der cdk oder von der Bindung eines der vielzähligen endogenen Inhibitorproteine herrühren.
- In der G1-Phase binden Cycline des D-Typs an mehrere verschiedene cdks, einschließlich cdk2, cdk4, cdk5 und cdk6, am häufigsten sind sie jedoch mit cdk4 und cdk6 assoziiert. Man nimmt an, dass Cycline des D-Typs als Wachstumsfaktorsensoren wirken, welche das Fortschreiten des Zellzyklus mit externen Signalstoffen verbinden. Cyclin E-cdk2-Komplexe erscheinen im Zellzyklus von Säugetieren nach den Cyclin-cdk-Komplexen des D-Typs. Die Synthese von Cyclin E wird streng kontrolliert und sie findet in der späten G1-Phase und der frühen S-Phase statt. Der Cyclin E-cdk2-Komplex ist wesentlich, damit die Zelle mit der DNA-Replikation beginnt.
- Die G1-Cycline, Cyclin D und Cyclin E, sind vorübergehend erzeugte Proteine, mit einer Halbwertszeit von etwa 20 Minuten. Man nimmt an, dass die kurze Halbwertszeit auf die PEST-Sequenz in den C-terminalen Regionen dieser Proteine zurückzuführen ist, deren Abbau entsprechend dem allgemeinen Pathway vermittelt zu sein scheint.
- Die G2-Cycline, Cyclin A und Cyclin B, sind während der Interphase stabil und sie werden besonders in der Mitosephase des allgemeinen Pathways abgebaut. Sowohl Cyclin A als auch Cyclin B scheinen nur abgebaut zu werden, wenn sie mit ihren cdk-Partnern komplexiert sind [CyclinA-cdk2 und Cyclin A/B-cdk1(cdk2)]. Der Cyclin B1-Abbau ist jedoch mit der Integrität des Mitose-Apparats am Ende der Metaphase verbunden. Ist die Spindel fehlerhaft zusammengesetzt, oder sind die Chromosomen fehlerhaft ausgerichtet, dann wird der Abbau von Cyclin B1 verhindert.
- Das Retinablastom-Protein (Rb), ein nukleares 105 kDa-Phosphoprotein, ist ein Substrat der Cyclin-cdk-Komplexe der cdks 2, 4 und 6 in der G1-Phase und es wirkt als einer der wichtigsten Kontrollpunkte im Zellzyklus über die sorgfältig abgestimmte Phosphorylierung und Dephosphorylierung. In den G0/G1-Phasen liegt Rb im hyperphosphorylierten Zustand vor. In dem Maße, in dem die Zelle in die späte G1-Phase eintritt, wird Rb durch D-Cyclin-Komplexe hyperphosphoryliert, welche Rb inaktivieren und die Zelle in die S-Phase voran treiben, was zum Fortschreiten des Zellzyklus und zur Zellteilung führt. Dieser Zustand der Hyperphosphorylierung von Rb dauert während der G2-Phase an. Während der späten M-Phase wird Rb dephosphoryliert, wodurch es in den hyperphosphorylierten Zustand zurück kehrt. Die Phosphorylierung des Rb-Proteins verändert seine Bindungscharakteristika; im hyperphosphorylierten Zustand bindet Rb an und sondert spezifische Transkriptionsfaktoren, wie E2F, ab, deren Bindung das Verlassen der G1-Phase verhindert. Wenn die cdks Rb hyperphosphorylieren, werden die Transkriptionsfaktoren freigesetzt, die dann die Transkription der Gene aktivieren, die zum Fortschreiten der S-Phase erforderlich sind, beispielsweise Thymidin-Kinase, myc, myb, Dihydrofolat-Reduktase und DNA-Polymerase-α.
- Ausgehend von der Rolle jedes Komplexes innerhalb des Pathways kann über die Lokalisation der Cyclin-cdk-Komplexe gleichfalls nur spekuliert werden. Die nuklearen Cycline A und E binden an p107 und p130, möglicherweise da sie sich im Kern befinden. Das Cyclin B1 von Säugetieren wird im Cytoplasma in der G2-Phase angereichert und tritt am Beginn der Mitose in den Kern ein. Das Cyclin B geht mit dem Spindelapparat einher, insbesondere mit den Spindelpolen, und man nimmt an, dass die Cyclin B-cdk2-Kinase an der Bildung der Spindel durch die Phosphorylierung der Bestandteile des Mitose-Apparats beteiligt ist. Zudem ist Cyclin B1 Teil eines Rückkopplungsmechanismus, der die richtige Anordnung des Mitose-Apparats der Metaphase sicherstellt. Humanes Cyclin B ist nahezu ausschließ lich mit dem Membranbestandteil assoziiert und insbesondere mit dem Golgi-Apparat. Cyclin B2-cdc2 ist an dem Abbau des Golgi-Apparats beteiligt, wenn die Zellen in die Mitosephase eintreten.
- Cdc2-Cyclin B-Kinase ist ein Schlüsselfaktor der Mitose und sie scheint stark konserviert zu sein und man nimmt an, dass sie an die Zellzyklus-Übergängen in allen eukaryontischen Zellen beteiligt ist. Histon H1 ist ein Substrat für cdc2-Cyclin B; Histon H1 ist in der Mitosephase an spezifischen Stellen selektiv phosphoryliert, man nimmt an, dass dies für die Chromatin-Kondensation von Bedeutung ist. Der cdc2-Cyclin B-Komplex phosphoryliert auch Lamin, das für die Aufspaltung der nuklearen Lamina verantwortlich ist. Die nukleare Lamina besteht aus einem Polymer von Lamin-Subeinheiten, die in der Mitosephase hyperphosphoryliert sind, und diese Phosphorylierung ist für ihren Abbau verantwortlich. Die Lamine sind Teil der intermediären Filament-Familie von Proteinen, und cdc2-Cyclin B phosphoryliert eine Reihe der Stellen, die in der Mitosephase an den cytoplasmatischen intermediären Filament-Subeinheiten phosphoryliert sind, und Vimentin und Desmin. Somit ist der cdc2-Cyclin B-Komplex an der Umgestaltung der Zellarchitektur in der Mitosephase beteiligt.
- Zudem ist cdc2-Cyclin B an der Umgestaltung der Mikrofilamente beteiligt, über die Phosphorylierung von nicht-muskulären Caldesmon, einem 83 kDa Protein, das an Actin und Calmodulin bindet und die Actomyosin ATPase-Aktivität hemmt. In der Mitosephase wird Caldesmon durch cdc2-Cyclin B phosphoryliert, was seine Affinität für Actin abschwächt und seine Dissoziation von den Mikrofilamenten bewirkt.
- Cdc2-Cyclin B ist an der Regulation des Actomyosin-Filaments beteiligt, indem das Myosin in dem kontraktilen Ring phosphoryliert wird, was die Zelle in zwei Zellen teilt (Cytokinese). In der Metaphase wird die regulatorische leichte Myosin II-Kette (MLC) an zwei wichtigen Stellen des N-terminalen Endes phosphoryliert. Ist das Myosin einmal phosphoryliert, wird es an der Wechselwirkung mit Actin gehindert. In der Anaphase werden diese zwei Stellen dephosphoryliert.
- Cdc2-Cyclin B spielt auch eine Rolle bei der Umgestaltung des Membranbestandteils in der Mitosephase. Cdc2-Cyclin B phosphoryliert beispielsweise rab1Ap und rap4p. Wird rab4p durch cdc2-Cyclin B phosphoryliert, dissoziiert es von dem Membranbestandteil.
- In der Mitosephase sind die meisten Formen der Transkription gehemmt. Wieder spielt cdc2-Cyclin B eine Rolle bei der Hemmung der pol III-vermittelten Transkription indem TFIIIB phosphoryliert wird. Nimmt man an, dass es für die pol I-, pol II- und pol III-vermittelte Transkription mehrere gemeinsame allgemeine Faktoren gibt, wie das TATA-bindende Protein (TBA), ist es wahrscheinlich, dass cdc2-Cyclin B an der Niederregulation aller Formen der Transkription in der Mitosephase beteiligt ist.
- Nimmt man an, dass Cyclin/cdK-Komplexe für die Auslösung des Zyklus der Zellteilung von Bedeutung sind, dann werden sie durch einen strengen Mechanismus reguliert. Seit ihrer ursprünglichen Entdeckung ist gezeigt worden, dass Cycline und cdks mit anderen Transkriptionsfaktoren und Proteinen in Wechselwirkung treten, die in einem breiten Umfang an den zellulären Pathways beteiligt sind. Cdk7 ist als ein Bestandteil in dem Transkriptionsfaktor IIH (TFIIH) identifiziert worden, der die Kinaseaktivität der RNA-Polymerase II in der C-terminalen Domäne (CTD) enthält. Vor kurzem ist gefunden worden, dass cdk8, die Partner von Cyclin C ist, auch die CTD von RNA-Polymerase II phosphoryliert, dass sie jedoch anscheinend keine CAK-Aktivität besitzt. Es ist somit augenscheinlich, dass die cdks zusätzlich zur Regulation des Zellzyklus in breitem Umfang an zellulären Funktionen teilhaben. cdk-Inhibitorproteine (CDIs) sind kleine Proteine, die spezifisch Cyclin-cdk-Komplexe binden und inaktivieren, oder monomere cdks. Diese Inhibitoren können basierend auf ihrer Sequenz und ihren funktionellen Ähnlichkeiten in zwei Familien eingeteilt werden. Die INK4-Familie schließt p15INK4B, p16INK4, p18 und p19 ein, die spezifisch cdk4 und cdk6 binden. p16INK4 und p15INK4B enthalten vier sich wiederholende Ankyrin-Einheiten und zusätzlich dazu, dass ihnen eine wesentliche Homologie gemeinsam ist, werden sie durch benachbarte Gene an dem 9p12-Locus codiert.
- Hohe zelluläre Spiegel an p16 führen zur Inaktivierung von cdk4, da p16 Cyclin D-cdk4- und Cyclin D-cdk6-Komplexe bindet. Es ist festgestellt worden, dass das Gen für p16INK4 (MTS1) ein potenzielles Tumorsuppressor-Gen ist, da es in einer Vielzahl von Tumorzelllinien und auch in einigen primären Tumoren umgeordnet, deletiert oder mutiert vorliegt. In einer Studie über erbliche Melanome wiesen etwa die Hälfte aller Familien Keimbahn-Mutationen in dem p16INK4-Gen auf. Rb ist ein Repressor von p16INK4. Die Inaktivierung von zellulärem Rb, entweder durch Mutation oder virale Antigene, korreliert mit ansteigenden Spiegeln an p16INK4. p16INK4, p15INK4B und p18 hemmen die Bindung von Cyclin D mit cdk4 und cdk6.
- Die zweite Familie von CDIs ist die Kip/Cip-Familie, die p21Cip1,WAF-1, p27Kip1 und p57Kip2 einschließt. p27KIP1 liegt in proliferierenden Zellen in latenter oder maskierter Form vor. Nach der Stimulierung ist p27KIP1 unmaskiert und bindet an Cyclin-cdk4/6-Komplexe und hemmt diese. Die Proteine der Kip/Cip-Familie weisen eine strenge Homologie in der N-terminalen Region auf, die Region, die an Cyclin-cdk-Komplexe bindet. Die Proteine der Kip/Cip-Familie binden vorzugsweise an und hemmen Cyclin-cdk-Komplexe, die in den Komplexen der G1- und S-Phase beteiligt sind, gegenüber denen die in der M-Phase beteiligt sind.
- p21 (auch bekannt als WAF1, Cip1 und Sdi1) wird durch p53 induziert und bildet einen ternären Komplex mit dem proliferierenden nuklearen Zellantigen (PCNA), einer Subeinheit von DNA-Polymerase δ in mehreren Cyclin-cdk2-Komplexen, einschließlich der Cycline A, D1 und E. Die p21WAF-1-Expression in wachsenden, ruhenden und alternden Zellen korreliert mit einer Rolle als ein negativer Regulator des Eintritts in die S- Phase. Die p21WAF-1-mRNA wird in dem Maße hoch reguliert, indem die Zellen alternde oder ruhende Zellen werden, und nach der Stimulierung von ruhenden Zellen mit Serum, und sie nimmt in dem Maße ab, indem die Zellen in die S-Phase eintreten. p21 inaktiviert Cyclin E-cdk2, Cyclin A-cdk2, und Cyclin D1-, D2- und D3-cdk4-Komplexe.
- Die Genanalyse einer Vielzahl humaner Tumore offenbarte eine unverhältnismäßige Anzahl veränderter Zellzyklus-Proteine und es ist diese Abweichung, von der man annimmt, dass sie einen abnormalen Zellzyklus bewirkt. Cyclin D1 ist beispielsweise das bcl-1/PRAD1-Proto-Onkogen, das in einer Vielzahl von humanen Tumoren entweder überexprimiert oder dereguliert wird. Das Cyclin D1/CCND1-Gen, das sich auf dem Chromosom 11q13 befindet, wird in einer Vielzahl von Krebserkrankungen verstärkt, hauptsächlich bei Brustkrebs nicht-kleinen Zell-Lungenkarzinomen. Dies korreliert mit der Beobachtung, dass die Überexpression von Cyclin D1 ein allgemeines Merkmal in den Tumoren mit diesem spezifischen 11q13-Amplicon ist. Das Gen für p16 ist in einer Vielzahl von Tumorzelllinien und in einigen primären Tumoren umgebaut, deletiert oder mutiert. Mutationen in cdk4, insbesondere eine Arg24Cys-Mutation, ist in zwei nicht miteinander verwandten Familien mit erblichen Melanomem identifiziert worden. Diese Mutation wurde bei 11/11 der Melanompatienten, 2/17 nicht befallenen und 0/5 Ehepartnern festgestellt (Zuo, L., et al., Nature Genetics, 12: 97–99, 1996). Diese Mutation hat eine spezifische Wirkung auf die p16INK4a-Bindungsdomäne von cdk4, sie hat jedoch keinen Einfluss auf die Fähigkeit, an Cyclin D zu binden und eine wirksame Kinase zu bilden. Im Ergebnis dieser Mutation ist der Cyclin D/cdk4-Komplex gegenüber der normalen physiologischen Hemmung durch p16INK4a resistent. Andere Studien haben nachgewiesen, dass bei etwa der Hälfte aller familiär miteinander verwandten Melanompatienten eine Verbindung zu der Region des Chromosoms 9p21, die das p16INK4a-Gen enthält, besteht. Die identifizierten Typen der p16INK4a-Mutationen schließen eine nonsense-Mutation, Spli ce-Donor-Mutationen, eine nicht identifizierte Mutation, die die p16INK4a-Transkription verhindert und 3 missense-Mutanten, die nicht in der Lage sind, an cdk4 oder cdk6 zu binden, ein.
- Die Überexpression von cdk4 als Ergebnis der Gen-Verstärkung ist in einer Studie von 32 Gliom-Zelllinien identifiziert worden (He, J., et al., Cancer Res. 54: 5804–5807, 1994). Diese Veränderung wurde innerhalb der zehn Fälle mit intakten p16-Genen beobachtet. Die Genanalyse von Gliom-Zelllinien offenbarte, dass 24 der 32 Gliom-Zelllinien eine der beiden alternativen Gen-Veränderungen aufwiesen, wobei jede darauf hinwies, dass eine erhöhte cdk4-Kinaseaktivität für die Entwicklung von Tumoren der Glia von Bedeutung ist. Cdk4 befindet sich auf dem langen Arm des Chromosoms 12 und es ist festgestellt worden, dass es bei bestimmten Tumoren überexprimiert ist, infolge seiner Verstärkung als ein Bestandteil eines Amplicons, das andere relevante Gene einschließt, wie SAS oder MDM2. Alle vorstehenden Bedingungen führen zur Aktivierung von cdk4. Die Überexpression der Cycline B1 und E bei Leukämie und Festtumor-Zelllinien, als auch veränderte Muster der Cyclin E-Expression bei Brustkrebs sind ebenfalls beschrieben worden.
- Die zelluläre Hyperproliferation findet bei einer Vielzahl von Erkrankungen statt. Die häufigsten hyperproliferativen Erkrankungen sind Neoplasmen, die typischerweise gemäß dem Ursprung des hyperproliferativen Gewebes bezeichnet werden. Neoplasmen werden als neues Wachstum von tierischem oder pflanzlichem Gewebe definiert, das dem Gewebe, in dem es entsteht, mehr oder weniger ähnelt, jedoch keine physiologische Funktion besitzt und sie sind gutartig, potenziell bösartig oder besitzen einen bösartigen Charakter. Neoplasmen entstehen im Ergebnis des Verlusts der normalen Kontrolle, was zu einem unreguliertem Wachstum führt. Neoplastische Zellen weisen keine Differenzierung auf und sie erwerben die Fähigkeit, in lokale Gewebe einzudringen und zu metastasieren. Neoplasmen können sich in jedem Gewebetyp jedes Organs in jedem Alter entwi ckeln. Das Auftreten und die Mortalitätsrate der Neoplasmen steigt im Allgemeinen mit dem Alter an, wobei bestimmte Neoplasmen häufig im Alter zwischen 60 und 80 auftreten (z. B. Prostata, Magen und Kolon). Andere Neoplasmen treten jedoch häufig im Alter zwischen der Geburt und dem 10. Lebensjahr auf (z. B. akute lymphoblastische Leukämie). Ernährung, die Einwirkung von Karzinogenen, insbesondere der Konsum von Tabak, und familiäre Vorbelastungen beeinflussen gleichfalls das Auftreten einzelner Neoplasmen.
- Neoplastische Zellen unterscheiden sich von normalen Zellen hinsichtlich einer Vielzahl bedeutender Aspekte, einschließlich des Verlusts an Differenzierung, einer erhöhten Anfälligkeit und einer verminderten Empfindlichkeit gegenüber Arzneistoffen. Ein anderer bedeutender Unterschied ist das unkontrollierte Wachstum von Zellen, von dem man annimmt, dass es aus dem Verlust des normalen zellulären Kontrollmechanismus dieser Zellen entsteht, er ist entweder desaktiviert, wird umgangen oder anderweitig nicht berücksichtigt, wodurch die neoplastischen Zellen ohne Bezug zu dem normalen Kontrollmechanismus proliferieren. Neoplasma ist eine abnormale Masse an Gewebe, dessen Wachstum das des normalen Gewebes übersteigt und das mit diesem nicht koordiniert wird, und das in der gleichen zu hohen Weise nach der Entfernung der Stimulatoren, die diese Veränderung bewirkten, andauert.
- Neoplasmen werden entweder als gutartig oder bösartig klassifiziert. Gutartige Neoplasmen zeigen ein langsames, lokalisiertes Wachstum, das üblicherweise entsprechend ihrer Verkapselung durch eine fibröse Bindegewebskapsel abgegrenzt wird. Während gutartige Neoplasmen selten den Tod des Organismus bewirken, weisen unbehandelte bösartige Neoplasmen eine hohe Wahrscheinlichkeit auf, den Organismus zu töten. Bösartige Neoplasmen sind im Allgemeinen nicht verkapselt und sie weisen üblicherweise eine schnellere Wachstumsgeschwindigkeit auf. Bösartige Neoplasmen dringen häufig in das umgebende Gewebe und Gefäße ein und breiten sich an entfernt liegenden Stellen des Gewebes aus. Maligne Neoplasmen werden generisch als „Krebs" oder als „Tumore" bezeichnet, wobei die letztere Bezeichnung Quellen bedeutet.
- Myeloproliferative Erkrankungen sind eine Gruppe von Krankheiten, die durch eine abnormale Proliferation einer oder mehrerer hämatopoetischen Zelllinien oder Bindegewebselemente charakterisiert sind. Vier Erkrankungen werden normalerweise in die myeloproliferativen Erkrankungen eingeschlossen: Polycythemia vera (primäre Polyzythemie; Vaquez-Syndrom), Myelofibrose (agnogenische myeloide Metaplasie), chronische myelogene Leukämie und primäre (essentielle) Thrombozythemie. Akute Leukämie, insbesondere Erythroleukämie, und paroxysmale nocternale Hämoglobinurie werden ebenfalls als myeloproliferative Erkrankungen klassifiziert. Jede dieser Krankheiten wird entsprechend ihrer vorherrschenden Merkmale oder entsprechend der Stellen der Proliferation identifiziert. Obwohl jede aus der Proliferation verschiedener Zellen entsteht, ist gezeigt worden, dass jede durch eine klonale Proliferation hervorgerufen wird, die auf dem Niveau einer pluripotenten Stammzelle entsteht, welches variierende Grade an abnormaler Proliferation von erythroiden, myeloiden und megakaryotischen Vorläufern im Knochenmark bewirkt. Alle myeloproliferierenden Erkrankungen weisen eine Tendenz auf, in akuter Leukämie zu enden.
- Leukämien sind bösartige Neoplasmen der blutbildenden Gewebe. Von mindestens zwei Viren ist bekannt, dass sie im Menschen Leukämie hervorrufen. Der Epstein-Barr-Virus geht mit dem Burkitt-Lymphom einher und der humane lymphotrophe T-Zell-Virus, der auch als humaner akuter Leukämie/Lymphom-Virus (HTLV-1) bezeichnet wird, ist mit einigen T-Zell-Leukämien und -Lymphomen in Verbindung gebracht worden. Die Einwirkung, insbesondere die längere Einwirkung chemischer Mittel, wie Benzol und einiger antineoplastischer Mittel, oder von ionisierender Strahlung, genetische Vorbelastung (z. B. Down-Syndrom) und einige familiäre Erkrankungen (z. B. Fanconi-Anämie) führen zu einer Vorbelastung gegenüber Leukämien.
- Die Entwicklung von Leukämien scheint während eines einzelnen Zellzyklus in einem oder mehreren Schritten mit nachfolgender Proliferation und klonaler Ausdehnung stattzufinden. Leukämien werden in neuerer Zeit gemäß ihrer zellulären Reife klassifiziert; akute Leukämien sind vorrangig undifferenzierte Zellpopulationen und chronische Leukämien sind reifere Zellformen. Akute Leukämien werden des weiteren in lymphoblastische (ALL, auch als akute lymphozytische Leukämie bekannt) und myeloide (AML, auch als akute myelozytische, myelogene, myeloblastische, myelomonoblastische Leukämie bekannt) Typen eingeteilt. Sie können weiter anhand ihrer morphologischen und cytochemischen Erscheinungsformen gemäß der Französisch-Amerikanisch-Britischen-(FAB)-Klassifikation oder gemäß ihrem Differenzierungstyp oder -grad klassifiziert werden. Chronische Leukämien werden entweder als lymphozytisch (CLL) oder myelozytisch (CML) klassifiziert. CLL ist durch das Auftreten reifer Lymphozyten im Blut, Knochenmark oder den lymphoiden Organen charakterisiert. CML ist durch das vorrangige Auftreten granulozytischer Zellen aller Differenzierungsstadien im Blut, Knochenmark, Leber, Milz und anderen Organen charakterisiert.
- Das myelodysplastische Syndrom (MDS) wird als eine chronische klonale proliferative Erkrankung charakterisiert, in der normales oder hyperzelluläres Knochenmark mit Anämie und Dysmyelopoese einhergeht. Hämatopoetische Zellen, die proliferieren können, schließen erythroide, myeloide und megakaryozytische Formen ein. MDS ist eine verhältnismäßig neue Bezeichnung für eine Gruppe von Krankheiten, die als Preleukämie, refraktorische Anämie, Ph-Chromosomen-negative chronische myelozytische Leukämie, chronische myelomonozytische Leukämie und agnogenische myeloide Metaplasie bekannt sind. Das FAB-System liefert eine weitere Klassifikation der Myelofibrose.
- Lymphome sind eine heterogene Gruppe von Neoplasmen, die in retikuloendothelialen und lymphatischen Systemen entstehen. Die wichtigsten Lymphom-Typen sind die Hodgkinsche Krankheit und das Nicht-Hodgkin-Lymphom, als auch das seltenere Burkitt-Lymphom und Mycosis fungoides. Die Hodgkinsche Krankheit ist eine chronische Erkrankung mit lymphoretikulärer Proliferation unbekannter Ursache, die in lokalisierter oder disseminierter Form vorliegen kann, und sie wird gemäß vier histopathologischer Profile klassifiziert. Die Nicht-Hodgkin-Lymphome sind eine heterogene Gruppe von Erkrankungen, bestehend aus neoplastischer Proliferation lymphoider Zellen, die üblicherweise im Körper disseminieren. Die füheren Begriffe, Lymphosarkom und Retikulum-Zellsarkom, werden jetzt durch Begriffe ersetzt, die die Herkunft der Zellen und die Biologie der Krankheit widerspiegeln. Die Rappaport-Klassifikation basiert auf der Histopathologie; auf dem Differenzierungsgrad des Tumors; und ob das Wachstumsmuster diffus oder nodulär ist. Die Lukes- und Collins-Klassifikation basiert auf der Herkunft der Zellen, insbesondere ob sie sich von T- oder B-Zellen ableiten, ob sie histiozytischer (oder monozytischer) Herkunft sind oder unklassifiziert. Die International Panel Working Formulation des Nationalen Krebsinstituts teilt die Nicht-Hodgkin-Lymphome unter Verwendung der vorstehenden Klassifikationen ein.
- Das Burkitt-Lymphom ist ein stark undifferenziertes B-Zell-Lymphom, das dazu tendiert, andere Stellen als die Lymphknoten oder das retikulendotheliale System zu befallen. Das Burkitt-Lymphom weist ähnlich wie andere Lymphome eine spezifische geografische Verteilung auf, die einen nicht identifizierten Insektenvektor und ein infektiöses Mittel vermuten lässt. Der Nachweis weist auf das Herpes-ähnliche Epstein-Barr-Virus hin.
- Mycosis fungoides ist ein nicht häufig vorkommendes chronisches T-Zell-Lymphom, das primär die Haut und manchmal innere Organe befällt.
- Plasmazelldyskrasien (PCDs), oder monoklonale Gammopathie, sind Erkrankungen, die durch unverhältnismäßige Proliferation eines Klons von Zellen charakterisiert sind, die nor malerweise für die Immunglobulin-(Ig)-Synthese verantwortlich sind, und durch die Anwesenheit eines/einer strukturell und elektrophoretisch homogenen IG's oder Polypetideinheit im Serum oder Urin. Die Krankheiten können vorrangig asymptomatisch hinsichtlich fortschreitender, offener Neoplasmen sein (z. B. multiples Myelom). Die Krankheit entsteht durch die unverhältnismäßige Proliferation eines Klons, der ein spezifisches Ig erzeugt: IgG, IgM, IgA, IgD oder IgE.
- Das multiple Myelom, auch als Plasmazellmyelom oder Myelomatose bekannt, ist eine fortschreitende neoplastische Erkrankung, die durch Plasmazelltumore des Knochenmarks und Überproduktion an einem intakten monoklonalen Ig (IgG, IgA, IgD oder IgE) oder Bence-Jones-Protein charakterisiert ist, das frei an monoklonalen leichten κ- oder λ-Ketten ist. Diffuse Osteoporose oder diskrete osteolytische Verletzungen entstehen aus der Umgestaltung durch expandierende Plasmazelltumore oder durch einen Osteoklasten-aktivierenden Faktor, der durch bösartige Plasmazellen sekretiert wird.
- Makroglobulinämie, oder primäre oder Waldenstrom-Makroglobulinämie, ist eine Plasmazelldyskrasie, an der B-Zellen beteiligt sind, die normalerweise IgM synthetisieren oder sekretieren. Makroglobulinämie unterscheidet sich vom Myelom oder anderen PCDs, und ist eine lymphomatöse Erkrankung. Viele Patienten weisen Symptome wie Hyperviskosität, Erschöpfung, Schwäche, Blutungen der Haut oder Schleimhaut, und so weiter auf.
- Störungen der schweren Ketten sind neoplastische Plasmazelldyskrasien, die durch Überproduktion an homogenen schweren γ-, α-, μ-, und δ-Ig-Ketten charakterisiert sind. Diese Störungen führen zu unvollständigen monoklonalen Igs. Das klinische Bild ähnelt mehr dem Lymphom als dem multiplen Myelom.
- Hypersplenie ist ein Syndrom, in dem zirkulierende Zytopenie mit Splenomegalie einhergeht. Die Behandlung von Patienten mit Hypersplenie erfordert eine Therapie der zugrunde liegenden Erkrankung, keine Milzektomie. Lymphoproliferative und myeloproliferative Erkrankungen sind manchmal, jedoch nicht allein, die Gründe für Hypersplenie. Myeloproliferative Erkrankungen bewirken Hypersplenie, einschließlich Polycythemie vera, Myelofibrose mit myeloider Metaplasie, chronischer myelogener Leukämie und im Wesentlichem Thrombozythemie. Chronische lymphozytische Leukämie und die Lymphome (einschließlich der Hodgkinschen Krankheit) sind spezifische hyperproliferative Erkrankungen, die Hypersplenie hervorrufen können.
- Lungengewebe ist die Stelle sowohl für gutartige als auch für bösartige primäre Tumore, als auch die Stelle der Metastasierung von Krebs vieler anderer Organe und Gewebe. Zigarettenrauchen bewirkt einen hohen prozentualen Anteil an Lungenkrebs, wie es in über neunzig Prozent der Fälle bei Männern und etwa siebzig Prozent der Fälle bei Frauen nachgewiesen wurde. Die Einwirkung von Mitteln aus dem beruflichen Umfeld, wie Asbest, Strahlung, Arsen, Chromate, Nickel, Chlormethylether, Giftgas und Hochofen-Emissionen, geht ebenfalls mit Lungenkrebs einher. Die häufigsten Typen an Lungenkrebs sind Akanthome, kleinzellige und großzellige Karzinome und Adenokarzinome.
- Etwa fünfundneunzig Prozent an Magenkrebs sind Karzinome; weniger häufig sind Lymphome und Leiomysarkome. Magenkarzinome werden gemäß ihrem wesentlichen Auftreten klassifiziert; protrudierend, penetrierend (der Tumor besitzt eine scharfe, gut ausgebildete Grenze und kann ulzeriert sein) und streuend oder sonstig, d. h. die Charakteristika von zwei der anderen Typen aufweisend sein.
- Pankreatische Krebsarten können exokrine Karzinome sein, die häufig Adenokarzinome sind, die eher aus den Zellen des Darmtrakts entstehen als aus Azinuszellen, oder endokrine Tumore, die Insuliome einschließen. Gastrin-produzierende pankreatische Tumore, die Zellen des nicht-β-Typs oder der duodenalen Darmwand einschließen, können das Zollinger-Ellison-Syndrom hervorrufen, ein Syndrom, das durch Hypergastrinämie gekennzeichnet ist. Manchmal rufen andere endokrine Abnormali täten, insbesondere der parathroiden, oder hypophysären und adrenalen Drüsen eine polyglanduläre Störung hervor, die als multiple endokrine Neoplasie (MEN) bekannt ist. Nicht-β-Inselzelltumore können ein Syndrom hervorrufen, das als Vipoma-Syndrom bekannt ist, welches durch eine lang anhaltende massive Wasserdiarrhöe charakterisiert ist.
- Neoplasmen des Darmtrakts schließen Tumore des Dünndarms, Tumore des Dickdarms, und Krebs von Kolon und Rektum ein. Gutartige Dünndarmtumore können aus jejunalen und ilealen Neoplasmen entstehen, die Leiomyome, Lipome, Neurofibrome und Fibrome einschließen. Bösartige Dünndarmtumore, wie Adenokarzinome, sind nicht häufig, und typischerweise entstehen sie in dem proximalen Jejunum. Patienten mit der Crohn-Erkrankung des Dünndarms sind für derartige Adenokarzinome eher anfällig als Patienten mit der Crohn-Erkrankung des Kolon. Bei Patienten mit der Crohn-Erkrankung neigen die Tumore zur distalen Verteilung in den umgangenen oder entzündeten Schlaufen des Darmtrakts. Karzinoide Tumore entstehen typischerweise im Dünndarm, insbesondere in dem Ileum, und in etwa der Hälfte der Fälle treten multiple Tumore auf. Das Kaposi-Syndrom, das häufig bei Empfängern von Transplantaten und AIDS-Patienten auftritt, weist in etwa der Hälfte der Fälle eine gastrointestinale Beteiligung auf. Die Verletzungen können überall in dem GI-Trakt auftreten, sie werden üblicherweise jedoch im Magen, dem Dünndarm oder dem distalen Kolon gefunden.
- Tumore des Dickdarms schließen Polypen des Kolons und Rektums ein. Polypen sind eine Gewebemasse, die in den Darmwänden des Kolons entsteht und in das Lumen protrudiert. Polypen werden auf der Grundlage ihrer Histologie als tubuläre Adenome, tubulovillöse Adenome, villöse Adenome, hyperplastische Polypen, Hamartome, juvenile Polypen, polypoide Karzinome, Pseudopolypen, Lipome, Leiomyome und sogar seltenen Tumore klassifiziert.
- Bösartige Tumore können auch in dem Anorektum entstehen. Diese sind epidermoide (Akanthom bzw. squamöse Zellen) Karzinome des Anorektums, die etwa drei der fünf Prozent der rektalen und analen Krebsarten umfassen.
- In westlichen Ländern stehen Kolon- und Rektumkrebs an zweiter Stelle nach dem Lungenkrebs, wobei in jedem Jahr mehr neue Fälle auftreten. In den USA starben 1989 etwa 75 000 Menschen an diesen Krebsarten; etwa 70% davon traten im Rektum und sigmoiden Kolon auf, und 95% davon waren Adenokarzinome.
- Neoplasmen der Leber schließen gutartige Neoplasmen, die verhältnismäßig häufig sind, jedoch oft nicht entdeckt werden, und bösartige Neoplasmen ein. Das hepatozelluläre Adenom ist das wichtigste gutartige Neoplasma der Leber. Asymptomatische kleine Hämangiome kommen bei ein bis fünf Prozent der Erwachsenen vor. Adenome des Gallengangs und andere mesenchymale Neoplasmen treten gleichfalls auf, sie sind jedoch verhältnismäßig selten. Bösartige Neoplasmen der Leber sind die häufigste Form hepatischer Tumore, und die Leber ist eine häufige Stelle für Metastasen, deren Verteilung über das Blut erfolgt, üblicherweise aus primären Tumoren der Lunge, der Brust, dem Kolon, dem Pankreas und dem Magen. Das Auftreten hepatozellulärer Karzinome geht in bestimmten Teilen Afrikas und Südostasiens mit dem chronischen Hepatitis-B-Virus einher. In Nordamerika, Europa und anderen Gebieten mit einer geringen Prävalenz ist der größte Teil der Patienten an einer Zirrhose erkrankt. Das fibrolamellare Karzinom ist eine andere Variante des hepatozellulären Karzinoms, das die charakteristische Morphologie der in das lamellare fibröse Gewebe eingedrungenen bösartigen Hepatozyten aufweist. Das fibrolamellare Karzinom befällt üblicherweise verhältnismäßig junge Erwachsene, und es geht nicht mit einer bereits vorhandenen Zirrhose, chronischen Hepatitis-B-Virusinfektion oder anderen bekannten Risikofaktoren einher. Andere primäre bösartige Erkrankungen der Leber schließen Cholangiokarzinom (ein Tumor, der in dem intrahepatischen Gallenephitel entsteht), Hepatoblastom (das eines der häufigsten Fälle bei Jugendlichen ist) und Angiosarkom (das mit der industriellen Einwirkung von Vinylchlorid einhergeht) ein. Leukämie und verwandte Erkrankungen können hepatisches Gewebe einschließen, wobei sie das Ergebnis der Infiltration mit abnormalen Zellen sein können.
- Multiple endokrine Neoplasie-Syndrome (MEN) sind eine Gruppe genetisch unterschiedlicher familiärer Erkrankungen, die adenomatöse Hyperplasie und bösartige Tumorbildung in mehreren endokrinen Drüsen einschließen. Drei verschiedene Syndrome sind identifiziert worden. Typ I (MEN-I) wird durch Tumore der parathyroiden Drüsen, der pankreatischen Inselzellen und der Hypophyse charakterisiert. Typ II (MEN-II) wird durch das meduläre Karzinom in der Schilddrüse, Pheochromozytom und Hyperparathyroidismus charakterisiert. Typ III (MEN-III) wird durch multiple mukosale Neurome, meduläres Karzinom der Schilddrüse, und Pheochromozytome charakterisiert.
- Das karzinoide Syndrom wird üblicherweise durch intestinale karzinoide Tumore hervorgerufen, die zu hohe Menge an vasoaktiven Substanzen sekretieren, einschließlich Serotonin, Bradykinin, Histamin, Prostaglandine und Polypetidhormone. Abnormale Spiegel dieser Substanzen rufen eine Vielzahl von Symptomen hervor, häufig episodisch auftretendes kutanes Flushing, Cyanose, abdominale Krämpfe, Diarrhöe und valvuläre Herzerkrankungen.
- Neoplasmen der Knochen und Gelenke können gutartig oder bösartig sein. Gutartige Tumore des Knochens schließen Osteochondrosarkome (osteokartilaginäre Exostosen), die häufigsten gutartigen Knochentumore bei Kindern im Alter zwischen 10 und 20 Jahren, gutartige Chondrome (die innerhalb des Knochens lokalisiert sind), die am häufigsten bei Kindern und jungen Erwachsenen im Alter zwischen 10 und 30 Jahren auftreten, Chondroblastome (die in der Epiphyse entstehen), die selten, aber bei Kindern im Alter zwischen 10 und 20 Jahren häufig auftreten, Chondromyxofibrome, Osteoidosteom, Riesenzelltumore und fibromatöse Verletzungen ein. Primäre bösartige Tumore des Knochens schließen osteogenes Sarkom (Osteosarkom), welches der zweithäufigste primäre Knochentumor ist, Fibrosarkome, bösartiges fibröses Histiozytom, Chondrosarkome, mesenchymales Chondrosarkom, Ewing-Tumor (Ewing-Sarkom), bösartige Lymphome des Knochens, multiples Myelom, und den bösartigen Riesenzelltumor ein.
- Primäre Krebsarten anderer Gewebe können in das Knochengewebe metastasieren. Die häufigsten sind Karzinome, die in der Brust, Lunge, Prostata, Niere und der Schilddrüse entstehen.
- Die Neoplasmen des Zentralnervensystem (ZNS) werden im Allgemeinen gemäß dem Organ klassifiziert. Primäre interkraniale Neoplasmen werden in sechs Klassen unterteilt: Tumore von (1) dem Schädel; (2) der Hirnhaut; (3) den kranialen Nerven; (4) der Neuroglia und des Ependyms; (5) der Epiphyse oder epiphysären Drüse; und (6) jene, die erblichen Ursprungs sind. Neoplasmen des Schädels schließen Osteom, Hämangiom, Granulom, Xanthom und Osteitis deformans ein. Die Neoplasmen der Hirnhaut schließen Mengiom, Sarkom und Glomatose ein. Die Neoplasmen der kranialen Nerven schließen Gliome des Sehnervs und Schwannome der 8. und 5. kranialen Nerven ein. Die Neoplasmen der Neuroglia schließen Gliome und Ependymome ein. Die Neoplasmen der Hypophyse oder des hypophysären Körpers schließen Hypophysen-Adenom und -Pinealom eine. Die Neoplasmen erblichen Ursprungs schließen Kraniopharyngiom, Chordom, Germiom, Teratom, dermoide Zyste, Agiom und Hämangioblastom ein.
- Neoplasmen des Rückenmarks sind Verletzungen, die das Rückenmark oder seine Wurzeln zusammendrücken, und die in dem Parenchym, den Wurzeln, der Hirnhaut oder den Rückenwirbeln entstehen. Primäre Neoplasmen des Rückenmarks sind weniger häufig als interkraniale Tumore. Metastasierende Verletzungen sind häufig und sie können aus Karzinomen der Lungen, der Brust, der Prostata, der Niere, der Schilddrüse oder aus Lymphomen entstehen.
- Neoplasmen der Geschlechtsorgane treten in jedem Alter und in beiden Geschlechtern auf; sie machen jedoch etwa 30% der Krebsarten bei Männern und 4% der Krebsarten bei Frauen aus. Das Adenokarzinom der Prostata ist eine der wesentlichen bösartigen Erkrankungen bei Männern über 50 Jahren. Man nimmt an, dass das Adenokarzinom der Prostata mit dem Hormon in Zusammenhang steht und seine Pathologie ist typischerweise glandulär. Das Karzinom der Niere, das Adenokarzinom, stellt nur etwa ein bis zwei Prozent der Krebsarten bei Erwachsenen dar, die meisten festen Nierentumore sind jedoch bösartig. Der Wilm-Tumor, ein embryonales Adenomyosarkom der Nieren treten fötal auf und es wird häufig über mehrere Jahre nicht diagnostiziert. Neoplasmen der Harnblase und des Harnleiters sind histologisch ähnlich. Neoplasmen der Harnblase können durch bekannte Karzinogene der Harnwege, wie Anilinfarbstoffe, induziert werden, und der häufigste ist das vorübergehende Zellkarzinom, weniger häufig ist das Akanthom. Seltenere Karzinome der Geschlechtsorgane schließen Karzinome der Harnröhre und des Penis ein. Neoplasmen des Hodens sind eine der häufigsten festen bösartigen Erkrankungen bei Männern unter 30 Jahren. Die meisten testikulären Tumore entstehen aus der primordialen Keimzelle und sie werden gemäß dem daran beteiligten Zelltyp klassifiziert.
- Brustkrebs ist die häufigste Krebsart bei Frauen. In den USA besteht für Frauen aller Altersgruppen ein Risiko für die Entwicklung von Brustkrebs von etwa 10%, das Risiko, an dieser Erkrankung zu sterben, beträgt jedoch nur etwa 3,6%. Das Risiko steigt jedoch mit dem Alter, der familiären Vorbelastung mit Brustkrebs, der Einwirkung von Bestrahlung an, und sogar die Ernährungsweise wird als ein erhöhtes Risiko angesehen.
- Brustkrebs wird routinemäßig gemäß der Östrogen- und Progesteron-Rezeptor-Analyse typisiert. Etwa zwei Drittel der Patienten weisen Östrogen-Rezeptor positive (ER+) Brusttumore auf. Man nimmt an, dass Tumore, die Progesteron positiv sind, einen wirksamen Östrogenrezeptor aufweisen und das die Anwesenheit beider Rezeptoren eine größere Wahrscheinlichkeit für das Ansprechen auf die bevorzugte endokrine Behandlung lie fert, als die Anwesenheit nur eines der Rezeptoren. Die endokrine Therapie, üblicherweise mit Tamoxifen, wird bei Östrogen-Rezeptor positiven Tumoren bevorzugt. Östrogene und Androgene sind ebenfalls wirksam, jedoch infolge unerwünschter Nebenwirkung, die durch höhere Spiegel dieser Hormone induziert werden, weniger bevorzugt als andere Formen der endokrinen Behandlung. Brustkrebs kann in nahezu jedes Organ des Körpers metastasieren, die häufigsten Stellen für Metastasen sind jedoch die Lunge, die Leber, der Knochen, die Lymphknoten und die Haut.
- Das lobuläre in situ Karzinom (LCIS) oder die lobuläre Neoplasie wird am häufigsten bei premenopausalen Frauen gefunden. Das duktale in situ Karzinom (DCIS) kommt sowohl bei pre- als auch bei postmenopausalen Frauen vor. DCIS bildet eine fühlbare Masse. LCIS und DCIS machen etwa 90% aller Brustkrebsarten aus. Die selteneren Formen, meduläre und tubuläre Verletzungen, haben eine etwas günstigere Prognose.
- Die häufigsten gynäkologischen Neoplasmen sind die endometrialen Karzinome, die an vierter Stelle nach Brustkrebs stehen, colorektaler und Lungenkrebs bei Frauen. Die endometrialen Karzinome werden anhand ihrer klinischen Stadien charakterisiert, ausgehend von dem in situ im Stadium 0 bis zur Metastasierung in entfernte Organe im Stadium IVB. Die endometrialen Karzinome produzieren typischerweise Östrogen und die neuesten Behandlungsverfahren sind die Operation und die Therapie mit Progesteron.
- Krebs der Ovarien macht etwa 18% aller gynäkologischen Neoplasmen aus. Etwa 80% der bösartigen Krebsarten der Ovarien entsteht in dem Ephitel der Ovarien und sie werden gemäß ihrer Histologie klassifiziert. Die Tumore können auch in den Keimzellen oder dem Stroma entstehen.
- Das Karzinom der Vulva macht etwa 3–4% aller gynäkologischen Neoplasmen aus. Vulväres Karzinom tritt im Allgemeinen nach der Menopause auf, und etwa 90% sind Akanthome. Etwa 4% sind Basalzellenkarzinome und der Rest schließt intraephiteli ale Karzinome, Adenokarzinom der Bartholin-Drüse, Fibrosarkom und Melanom ein.
- Das vaginale Karzinom macht etwa 1% aller gynäkologischen bösartigen Erkrankungen aus, wobei es am häufigsten im Alter zwischen 45 und 65 Jahren auftritt. Etwa 95% aller vaginalen Karzinome sind Akanthome. Das primäre Karzinom des Eileiters ist selten und typischerweise wird es direkt oder durch das lymphatische System verteilt.
- Trophoblastische Erkrankungen oder Neoplasmen trophoblastischen Ursprungs können einer intra- oder extrauterinen Schwangerschaft folgen. Eine degenerative Schwangerschaft führt zu einem Blasenmole, von dem etwa 80% gutartig sind.
- Neoplasmen können im Ohrkanal entstehen und das Hörvermögen beeinflussen. Ceruminome, die ebenfalls entstehen können, sind typischerweise bösartig, obwohl sie im histologischen Befund gutartig erscheinen und sie werden durch operative Entfernung behandelt. Karzinome Basalzellen und Akanthome entwickeln sich häufig am Außenohr infolge der normalen Sonneneinstrahlung und sie werden typischerweise ebenfalls operativ entfernt. Das Mittelohr könnte die Stelle für Akan-thome sein. Nicht-chromaffine Paragangliome können in den Schläfenbeinen entstehen.
- Der häufigste bösartige Tumor der Nase und des paranasalen Sinus ist das Akanthom, weniger häufig sind adenoide zystische und mucoepidermode Karzinome, bösartige Tumormischtypen, Adenokarzinome, Lymphome, Fibrosarkome, Osteosarkome, Chondrosarkome und Melanome.
- Das Akanthom der Nasopharynx wird häufig bei Kindern und jungen Erwachsenen beobachtet.
- Die häufigsten bösartigen Erkrankungen des oberen Respirationstrakts sind die Akanthome der Mandeln und des Kehlkopfs. Beide treten häufig bei Männern auf und sie gehen mit Tabakrauchen und Alkoholkonsum einher; etwa 85% der Patienten, die an Krebs des Kopf- oder Nackenbereichs erkranken, weisen eine Vorgeschichte von Alkohol- oder Tabakkonsum auf.
- Im Kopf- oder Nackenbereich sind etwa 90% der Krebsarten Akanthome (epidermoide Karzinome). Melanome, Lymphome und Sarkome sind verhältnismäßig seltene Formen primärer Krebsarten des Kopf- oder Nackenbereichs. Krebsarten des Kopf- und Nackenbereichs werden gemäß ihrer Größe und der Stelle, die in das primäre Neoplasma miteinbezogen ist; gemäß der Anzahl und Größe der Metastasen in den zervikalen Lymphknoten; und dem Nachweis entfernter Metastasen klassifiziert.
- Ophthomalogische Krebsarten können in der Haut der Augenlieder entstehen und sie können gutartig oder neoplastisch sein. Gutartig wachsende Krebse sind häufig Xantheplasmen, die subkutan gelb-weiße flache Plaques aus Lipidmaterial bilden. Basalzellenkarzinome sind häufiger; die Behandlung ist typischerweise die operative Entfernung oder die Bestrahlungstherapie. Andere weniger häufige bösartige Tumore sind Akanthome oder Drüsenkarzinome der Augenlider und andere Melanomtypen. Die häufigste primäre okulare bösartige Erkrankung ist das bösartige Melanom des Choroids.
- Tumore entstehen auch im Hautgewebe, und sie schließen sowohl gutartige Tumore, wie Mole, Lipome und ähnliche, als auch bösartige Tumore ein. Etwa 40–50% der bösartigen Tumore entstehen aus den Melanozyten der Mole. Bösartige Hautkrebsarten sind entweder Basalzellenkarzinome oder Akanthome und sie entstehen oft in den Hautgebieten, die der Sonne ausgesetzt sind. Sie sind die häufigsten bösartigen Erkrankungen und ihr Auftreten wächst. Weniger häufige bösartige Erkrankungen schließen das bösartige Melanom, die Paget-Erkrankung der Brustwarzen oder des östramammären Durchgangs; das Kaposi-Sarkom (KS) und das kutane T-Zell-Lymphom (Mycosis fungiodes) ein. Das Auftreten von KS steigt im Ergebnis des ansteigenden Auftretens von AIDS. KS entsteht bei etwa einem Drittel der Patienten mit AIDS.
- Orale Krebsarten machen etwa 5% der Krebsarten bei Männern und 2% der Krebsarten bei Frauen auf. Die häufigste Form des oralen Krebs ist das Akanthom. Das Auftreten steigt mit dem Alter und den Risikofaktoren an, insbesondere dem Tabak- und Alkoholkonsum.
- Die Operation ist die älteste wirksame Form der Behandlung von Neoplasmen. Der Erfolg wird zum großen Teil erreicht, wenn das Neoplasma in seinen frühen Stadien entdeckt wird und es nicht metastasiert hat. Die Bestrahlung ist ebenfalls eine wichtige Therapie, und sie ist die bevorzugte Therapie vieler Neoplasmen, wie die Hodgkinsche Krankheit, früher Nicht-Hodgkin-Lymphome und dem Akanthom des Kopf- und Nackenbereichs. Die Bestrahlung hat sich als Ergänzung zur Operation und antineoplastischen Arzneistoffen als sehr erfolgreich gezeigt.
- Antineoplastische Arzneistoffe sind gleichfalls zur Behandlung von Neoplasmen geeignet und sie werden gemäß ihrem Wirkmechanismus klassifiziert. Zahlreiche Kombinationen, typischerweise antineoplastische Arzneistoffe mit verschiedenen Wirkmechanismen, haben sich als eine besonders wirksame Therapie erwiesen, sie gestatten geringe Dosierungen und verringern oft negative Nebenwirkungen. Antineoplastische Arzneistoffe zielen oft auf grundlegende biologische Abläufe, die für die Zellreplikation oder das Zellwachstum notwendig sind.
- Alkylierungsmittel, wie Mechlorethamin und Cyclophosphamid alkylieren DNA und begrenzen die DNA-Replikation.
- Antimetaboliten, die auf die Zerstörung der notwendigen Wege der Zellteilung gerichtet sind, schließen ein:
- Folat-Antagonisten binden an Dehydrofolat-Reduktase und sie behindern die Pyrimidinsynthese. Folat-Antagonisten sind für die S-Phase spezifisch. Methotrexat ist ein sehr häufig verwendeter antineoplastischer Folat-Antagonist.
- Purin-Antagonisten blockieren de novo die Purinsynthese und sie sind für die S-Phase spezifisch. 6-Mercaptopurin ist ein Beispiel eines Purin-Antagonisten.
- Pyrimidin-Antagonisten behindern die Thymidylatsynthese, wobei die Produktion von Thymidin verringert wird und sie sind für die S-Phase spezifisch. Ein häufig verwendeter Pyrimidin-Antagonist ist 5-Fluoruracil.
- Cytarabin hemmt die DNA-Polymerase und es ist für die S-Phase spezifisch.
- Pflanzliche Alkaloide schließen Vinca-Alkaloide, wie Vinblastin und Vincristin, und Podophyllotoxine, wie Etopsid, ein. Pflanzliche Alkaloide sind in der Metaphase wirksam und sie hemmen die Mitose durch eine Vielzahl von Mechanismen, einschließlich der Veränderung der mikrotubulären Proteine.
- Antibiotika schließen Doxorubicin und Daunomycin ein, die sich zwischen die DNA-Stränge schieben, wodurch die Entknäulung der DNA gehemmt wird; Bleomycin, das Schnitte in den DNA-Strängen bewirkt; und Mitomycin, das die DNA-Synthese hemmt, indem es als ein bifunktioneller Alkylator wirkt.
- Nitrosoharnstoffe schließen Carmustin und Lomustin ein und sie alkylieren die DNA oder bewirken die Entstehung von Carbamoylat-Aminosäuren in Proteinen.
- Anorganische Ionen, wie Cisplatin, bewirken inter- und intra-Einschiebung von DNA-Strängen, wobei die Entknäulung von DNA gehemmt wird.
- Mittel, die die biologische Ansprechbarkeit modifizieren, wie Interferone, besitzen antiproliferative Wirkungen, ihre genaue Rolle ist jedoch nicht bekannt. Interferone schließen α-(Leukozyt)-Interferon, β-(Fibroblast)-Interferon und γ-(Lymphozyt)-Interferon ein.
- Enzyme, wie Asparaginase, werden ebenfalls zur Veränderung des metabolischen Wegs verwendet, der für die Krebszellen von Bedeutung ist. Asparaginase entzieht der Zelle Asparagin, von dem die Leukämie-Zellen abhängig sind.
- Hormone und ihre Analoge, wie Tamoxifen, Flutamid und Progesteron, weisen nicht-spezifische Wirkungen auf, sie sind jedoch zur Behandlung bestimmter Neoplasmen geeignet, von denen bekannt ist, dass sie auf Hormone ansprechen, insbesondere für Neoplasmen der Brust, Ovarien und Protasta. Tamoxifen, das häufig zur Behandlung von Neoplasmen der Brust verwendet wird, versetzt die Zellen in den Ruhezustand und bindet an den Östrogen-Rezeptor. Flutamid, das häufig zur Behandlung von Neoplasmaen der Prostata verwendet wird, bindet an den Androgen-Rezeptor.
- Cytokinine sind natürlich vorkommende und künstliche Regulatoren des Pflanzenwachstums. Natürliche Cytokinine scheinen nicht-spezifische Inhibitoren verschiedener Proteinkinasen zu sein. Die molekularen Mechanismen, über die die Cytokinine das Zellwachstum und die Zellteilung regulieren, müssen noch ermittelt werden. Studien haben darauf hingewiesen, dass Cytokinine die Zugänglichkeit zu dem DNA-Templat erhöhen, die RNA-Polymerase aktivieren, die Polyadenylierung und die Sekundärstruktur der mRNA beeinflussen und die Bildung und Aktivität der Polyribosomen stimulieren können. Man nimmt an, dass Cytokinine die Zellteilung beeinflussen, indem sie mit den Regulatorproteinen des Zellzyklus in Wechselwirkung treten. Sowohl Cytokinine als auch Cytokinin-abhängige Kinasen (cdks) wirken an multiplen und ähnlichen Kontrollpunkten des Zellzyklus, beispielsweise in den G1/S- und G2/M-Übergängen und den S- und M-Phasen.
- Olomoucin, 6-[(Benzylamino)-2-(2-hydroxyethyl)amino)]-9-methylpurin, wurde als erstes als Herbizid entdeckt. Kürzlich ist festgestellt worden, dass Olomoucin ein künstliches Cytokinin ist, das bei einer mikromolaren Konzentration spezifisch einige cdks hemmt, einschließlich p34cdc2/Cyclin B-Kinasen, es hat jedoch keine Wirkung auf andere wichtige Proteinkinasen, wie cAMP- und cGMP-abhängige Kinasen, und Proteinkinase C. Es ist kürzlich gezeigt worden, dass Olomoucin eine gute Selektivität für CDK-Cyclin-Proteinkinasen aufweist, dass es jedoch nur eine mittlere hemmende Wirkung besitzt, mit einem IC50 von etwa 7 μM. Vesely, J., et al., Eur. J. Biochem., 1994, 224, 771–786. Eine 2,4 A Kristallstruktur von Olomoucin, das mit cdk2 co-kristallisiert ist, offenbarte, dass der Purin-Teil von Olomoucin an die konservierte ATP-Bindungskassette bindet, während die Benzylaminogruppe in einen Be reich der aktiven Stelle ausgedehnt wird, die eindeutig auf die cdk-Kinasen zurückzuführen ist.
- WO97/16452 offenbart 2-Amino-6-anilin-purin-Derivate, die eine hemmende Wirkung auf p34cdc2/Cyclin Bcdc13-Kinase, aufweisen.
- Roscovitin, 2-(1-Ethyl-2-hydroxyethylamino)-6-benzylamino-9-isopropylpurin, ist ein kürzlich synthetisiertes Purin, von dem gezeigt worden ist, dass es Selektivität gegenüber einigen Cyclin-abhängigen Kinasen aufweist und dass es 10mal wirksamer auf cdk2 und cdc2 ist als Olomoucin (Meijer, L., et al., Eur. J. Biochem., 243: 257, 1997 und PCT/FR96/01905). Meijer et al. beschreiben, dass die meisten Kinasen durch Roscovitin nicht wesentlich gehemmt werden. Cdc2-Cyclin B, cdk2-Cyclin A, cdk2-Cyclin E und cdk5-p35 werden jedoch wesentlich mit IC50-Werten von 0,65, 0,7, 0,7 bzw. 0,2 μM gehemmt. Im Gegensatz dazu zeigte Roscovitin IC50-Werte, die für cdk4-Cyclin D1 und cdk6-Cyclin D2 größer sind als 100 μM.
- Havlicek, L., et al., J. Med. Chem. (1997) 40: 408–412 beschreiben, dass Roscovitin und verwandte Analoge, die in den 2-, 6- und/oder 9-Positionen substituiert sind, p34cdc2-Cyclin B-Kinasen hemmen. Keines der Analoge hatte ID50-Werte, die weit über dem des (R)-Enantiomers von Roscovitin lagen, welches einen ID50-Wert von 0,2 μM besaß. Das (S)-Enantiomer hatte einen ID50-Wert von 0,8 μM; das racemische Gemisch (R/S) hatte einen ID50-Wert von 0,65 μM. Diese Autoren schlussfolgerten, dass der N6-Benzyl-Substituent von Roscovitin im Vergleich zu den Isopentyl- oder Cyclohexylmethyl-Substituenten hervorragend war.
- Das Nationale Krebsinstitut (NCI) ist eine Organisation der US-Regierung, die auf das Auffinden und die Entwicklung neuartiger onkologischer Therapieprodukte gerichtet ist. 1985 etablierte das NCI eine neue Screening-Therapie für Krebs, die humane Tumorzelllinien in einem in vitro-Assay ebenso einschließt wie das Screening bezüglich des primären Tumors. Eine Gesamtheit von sechs humanen Tumorzelllinien, abgeleitet von sieben Krebstypen (Lunge, Kolon, Melanom, Blase, Ovarien, Gehirn und Leukämie) wurde für den Einschluss in das NCI-Projekt ausgewählt (Grever, M. R., et al., Seminars in Oncology, 19: 1992: 622–638). Die in diesen Assays verwendeten Protokolle sind ebenfalls in der Literatur beschrieben worden. Die American Type Tissue Collection (ATCC) dient der Hinterlegung für diese und andere Tumorzelllinien. Geeignete humane Tumorzelllinien schließen die nachfolgenden ein:
MCF7: humanes Brust-Adenokarzinom, Hormon-abhängig;
MDA-MB-231: humanes Brust-Adenokarzinom, Hormon-unabhängig;
HT-29: humanes Kolon-Adenokarzinom; mittlerer Differenzierungsgrad II;
HCT-15: humanes Kolon-Adenokarzinom;
A549: humanes nicht-kleinzelliges Lungenkarzinom;
DMS-114: humanes kleinzelliges Lungenkarzinom;
PC-3: humanes Prostata-Adenokarzinom; Hormon-unabhängig; und
DU 145: humanes Prostata-Adenokarzinom; Hormon-abhängig. - Shekan, P., et al., J. Natl. Cancer Inst. 82: 1107–1112, 1990, führten geeignete Protokolle für die Verwendung derartiger Tumorzelllinien zum Screening nach antineoplastischen Arzneistoffen fort.
- Meijer, et al., supra, beschreiben, dass Roscovitin die Proliferation in dem Krankheit-orientierten in vitro-Screening des NCI, d. h. 60 humane Tumorzelllinien, umfassend neun Tumortypen (Leukämie, nicht-kleinzelliger Lungenkrebs, Kolonkrebs, Krebs des zentralen Nervensystems, Melanom, Ovarienkrebs, Blasenkrebs, Prostatakrebs, Brustkrebs) mit einem mittleren IC50-Wert von 16 μM hemmt. Die Ergebnisse der individuellen Tumorlinien wurden nicht mitgeteilt.
- Zwei verschiedenen cdk-Inhibitoren, Flavopiridol und Olomoucin, unterdrücken den Tod der neuralen PC12-Zellen und sympathischen Neuronen in zwei Modellsystemen des neuronalen Überlebens (Park, et al., J. Biol. Chem. 271 (14): 8161–8169, 1996). Die Konzentration, die jeweils zur Förderung des Überlebens erforderlich ist, korreliert mit der Menge, die zur Hemmung der Proliferation erforderlich ist. Die neuronale Apoptosis (Apoptose) ist ein bedeutender Aspekt sowohl im Hinblick auf die Entwicklung des Nervensystems als auch als ein Bestandteil von neuronaler Verletzung und Krankheit.
- Die PC12-Zelllinie wurde ursprünglich von einem adrenal modulären Pheochromocytom der Ratte abgeleitet. Werden sie in Serum-enthaltendem Medium kultiviert, teilen sich die PC12-Zellen und bilden Vorläufer von adrenalen chromaffinen Zellen und sympathischen Neuronen. Die Zugabe von Nerven-Wachstumsfaktor (NGF) verleiht den PC12-Zellen phenotypische Eigenschaften von sympathischen Neuronen. Nach der Entfernung entweder des Serums oder von NGF unterliegen sowohl die naiven als auch die neuronal differenzierten PC12-Zellen der Apotose, die analog mit der Ansprechbarkeit der sympathischen Neuronen abläuft.
- Die Rolle der Zellregulation bei der Apoptose kann anhand der Entfernung von NGF oder des Serums, die zu einem ungeordneten Fortschreiten des Zellzyklus und zum Zelltod der naiven PC12-Zellen führen, veranschaulicht werden. Cdk-Inhibitoren hatten den Tod dieser Proliferations-kompetenten naiven PC12-Zellen nach der Entfernung des Oberflächenträgers nicht verhindert. Es wurde die Hypothese aufgestellt, dass post-mitotisch differenzierte oder sympathische Neuronen den Versuch eines ungeeigneten Wiedereintritts in den Zellzyklus nach der Entfernung von NGF unternehmen, der zum Zelltod führt. Die Einwirkung von Flavopiridol oder Olomoucin, die cdks hemmen, verhindert jedoch die Apoptose in diesen Zellen.
- Veränderungen in der Aktivität von cdks und Cyclinen werden während der Apoptose vieler verschiedener Zelltypen beobachtet. Die Camptothecin- oder araC-induzierte Apoptose von HL60-Zellen geht mit erhöhter cdc2-Aktivität und Cyclin E-assoziierten Kinaseaktivität einher. Die Camptothecin-induzierte Apoptose von RKO-Zellen geht mit einem Anstieg in der Expression von Cyclin D1 einher.
- Camptothecin bewirkt den apoptotischen Tod von cerebralen kortikalen Neuronen der Ratte. Morris und Geller, J. Cell Biol. 134: 757–770 (1996). Camptothecin-behandelte nicht proliferierende neuronal differenzierte PC12-Zellen sterben innerhalb von 6 Tagen nach der Behandlung, und kultivierte sympathische Neuronen der Ratte sterben innerhalb von 5 Tagen nach der Behandlung, sogar in Anwesenheit von NGF. Park et al. J. Neurosci. 17(4): 1256–1270 (1997). Jedoch die Verabreichung von Olomoucin oder Flavopiridol, sowohl beide als auch einzeln, in An- oder Abwesenheit von Camptothecin führte zu einem annähernd 30%igen Zelltod am Tag 6. Ein maximaler Schutz für PC12-Zellen, oder sympathische Neuronen der Ratte, gegen den Zelltod wurde mit 1 μM Flavopiridol und 200 μM Olomoucin beobachtet, welches die minimalen Konzentrationen sind, die die DNA-Synthese proliferierender PC12-Zellen vollständig hemmen. Die Verabreichung von iso-Olomoucin, einem inaktiven Analogen von Olomoucin, verhinderte den Zelltod von Camptothecin-behandelten neuronalen Zellen nicht.
- Es wurde gleichfalls gezeigt, dass Flavopiridol und Olomoucin vor dem Camptothecin-induzierten kortikalen neuronalen Tod schützen. Park et al., J. Neurosci. 17(4): 1256–1270 (1997). Die IC50-Werte von Flavopiridol und Olomoucin betrugen 0,1 μM bzw. 100 μM. Die Verabreichung von iso-Olomoucin verhinderte den Zelltod von Camptothecin-behandelten neuronalen Zellen nicht.
- Es gibt verschiedene Schlussfolgerungen aus den obenstehenden Beobachtungen. Es ist gut bekannt, dass bei Patienten, die mit Bestrahlung oder antineoplastichen Mitteln behandelt wurden, unerwünschte Nebenwirkung auftreten, einschließlich der Entwicklung neuer Neoplasmen oder einer unerwünschten zellulären Apoptose. Beispielsweise entwickeln einige Patienten, die wegen refraktorischer Leukämie mit einer hohen Dosis araC behandelt wurden, ein zerebrales Toxizitätssyndrom, das durch den Verlust von Purkinje-Neuronen charakterisiert ist. Winkelman und Hinges, Ann Neurol. 14: 520–527 (1983) und Vogel und Horouipian, Cancer 71: 1303–1308 (1993). Es ist beschrieben worden, dass Patienten, die mit Cisplatin behandelt wurden, periphere Neuropathien entwickeln. Wallach, et al., J. Fla. Med. Assoc. 79: 821–822 (992) und Mansfield und Castillo, AJNR Am. J. Neuroradiol. 15: 1178–1180 (1994). Im Hinblick auf diese Beobachtungen könnte entweder die co-Verabreichung oder die reine Verabreichung der Verbindungen der vorliegenden Erfindung zur Behandlung von Neoplasmen die zelluläre Apoptose verringern oder ausschließen, insbesondere die durch die Behandlung mit antineoplastischen Mitteln oder Bestrahlung hervorgerufene neuronale Schädigung.
- Cerebrovaskuläre Erkrankungen sind die häufigste Ursache von neurologischen Versehrtheiten in westlichen Ländern. Die hauptsächlichen spezifischen Typen cerebrovaskulärer Erkrankungen sind cerebrale Insuffizienz entsprechend der vorübergehenden Störungen des Blutfluss, Infarkt, Hämorrhagie und arteriovenöse Fehlbildung. Schlaganfall bezeichnet im Allgemeinen ischämische Verletzungen. Bei cerebrovaskulären Erkrankungen findet eine unerwünschte neuronale Apoptose statt. Die Behandlung mit Inhibitoren von cdks kann ein Verfahren sein, neuronale Schädigungen und Degenerationen in derartigen Fällen zu verhindern.
- Zusammenfassung der Erfindung
- Die vorliegende Erfindung liefert neuartige Verbindungen der Formel (I) worin R ausgewählt ist aus der Gruppe, bestehend aus R2, R2NH-, oder H2N-R3, worin
R2 ausgewählt ist aus der Gruppe, bestehend aus C1-C8-Alkyl und worin Z ausgewählt ist aus der Gruppe, bestehend aus Phenyl, einem Heterocyclus, ausgewählt aus Piperidinyl, Pyridinyl, Isoxazolyl, Tetrahydrofuranyl, Pyrrolidinyl, Morpholinyl, Piperazinyl, Benzimidazolyl, Thiazolyl, Thiophenyl, Furanyl, Indolyl, 1,3-Benzodioxolyl, Tetrahydropyranyl, Imidazolyl, Tetrahydrothiophenyl, Pyranyl, Dioxanyl, Pyrrolyl, Pyrimidinyl, Pyrazinyl, Triazinyl, Oxazolyl, Purinyl, Chinolinyl und Isochinolinyl, und Cycloalkyl, worin jedes R4 unabhängig Wasserstoff oder C1-C4-Alkyl darstellt, und n eine ganze Zahl von 1–8 ist;
worin jedes C1-C8-Alkyl und Z gegebenenfalls mit 1 bis 3 Substituenten substituiert ist, die gleich oder verschieden sein können und die ausgewählt sind aus der Gruppe, bestehend aus Hal, OH, und C1-C4-Alkyl;
R3 C1-C8-Alkylen darstellt; und
R1 ausgewählt ist aus der Gruppe, bestehend aus Cyclophenyl und Isopropyl, und die pharmazeutisch verträglichen Salze, optischen Isomere, und Hydrate davon. - Die vorliegende Erfindung stellt ein Verfahren zur Hemmung des Fortschreitens des Zellzyklus bereit. Genauer stellt die vorliegende Erfindung ein Verfahren zur Hemmung von cdk-2 bereit.
- Die vorliegende Erfindung stellt auch ein Verfahren zum Verhindern von Apoptose in neuronalen Zellen bereit. Ein besonders bevorzugtes Verfahren der vorliegenden Erfindung ist das Verhindern von Apoptose von neuronalen Zellen, die durch antineoplastische Mittel induziert wird oder die eine Folge der cerebrovaskularen Erkrankung ist. Eine andere bevorzugte Ausführungsform der vorliegenden Erfindung ist ein Verfahren zum Verhindern von Apoptose, die durch die Entfernung von Sau erstoff induziert wird. Eine bevorzugtere Ausführungsform der vorliegenden Erfindung stellt ein Verfahren zum Verhindern von Apoptose, die durch cerebrovaskuläre Erkrankungen induziert wird, bereit. Eine andere bevorzugte Ausführungsform der vorliegenden Erfindung stellt ein Verfahren zum Verhindern von Apoptose, die durch Schlaganfall oder Infarkt induziert wird, bereit.
- Die vorliegende Erfindung stellt ein Verfahren zur Hemmung der Entwicklung von Neoplasmen bereit. Die vorliegende Erfindung stellt ein Verfahren zur Behandlung eines Patienten, der von einem neoplastischen Krankheitszustand befallen ist, umfassend die Verabreichung einer Verbindung der bereitgestellten Formel, bereit. Es ist bevorzugt, dass die verabreichte Menge eine therapeutisch wirksame Menge einer Verbindung der Formel ist. Ein bevorzugtes Verfahren der vorliegenden Erfindung ist es, eine einzelne Verbindung der bereitgestellten Formel zu verabreichen. Alternativ ist es ein bevorzugtes Verfahren der vorliegenden Erfindung, eine Menge der Verbindung der Formel in Verbindung mit anderen antineoplastischen Mitteln zu verabreichen.
- Zudem stellt die vorliegende Erfindung eine Zusammensetzung bereit, umfassend eine nachweisbare Menge einer Verbindung der Formel (I) in Anmischung oder anderweitig in Verbindung mit einem inerten Träger. Die vorliegende Erfindung stellt auch eine pharmazeutische Zusammensetzung bereit, umfassend eine wirksame hemmende Menge einer Verbindung der Formel (I) in Verbindung mit einem oder mehreren pharmazeutisch verträglichen Trägern oder Exzipienten.
- Genauere Beschreibung der Erfindung
- Die vorliegende Erfindung stellt neuartige Verbindungen der Formel (I) bereit worin R ausgewählt ist aus der Gruppe, bestehend aus R2, R2NH-, oder H2N-R3-, worin
R2 ausgewählt ist aus der Gruppe, bestehend aus C1-C8-Alkyl und worin Z ausgewählt ist aus der Gruppe, bestehend aus Phenyl, einem Heterocyclus wie vorstehend definiert, und Cycloalkyl, jedes R4 unabhängig Wasserstoff oder C1-C4-Alkyl darstellt, und n eine ganze Zahl von 1–8 ist; worin jedes
C1-C8-Alkylen und Z, gegebenenfalls substituiert ist mit 1 bis 3 Substituenten, die gleich oder verschieden sein können, ausgewählt aus der Gruppe, bestehend aus Hal, OH, und C1-C4-Alkyl;
R3 C1-C8-Alkylen darstellt; und
R1 ausgewählt ist aus der Gruppe, bestehend aus Cyclopentyl und Isopropyl, und die pharmazeutisch verträglichen Salze, optischen Isomeren und Hydrate davon. - In den Bereich der Formel (I) fallen auch neuartige Verbindungen der Formel (Ia). worin R R2 darstellt, worin
R2 C1-C8-Alkyl, gegebenenfalls substituiert mit 1 bis 3 Substituenten, die gleich oder verschieden sein können, ausgewählt aus der Gruppe, bestehend aus Hal, OH, und C1-C4-Alkyl, darstellt;
R1 ausgewählt ist aus der Gruppe, bestehend aus Cyclopentyl und Isopropyl, und die pharmazeutisch verträglichen Salze, optischen Isomeren, und Hydrate davon. - Auch in den Bereich der Formel (I) fallen neuartige Verbindungen der Formel (Ib), worin R R2 darstellt,
R2 darstellt, worin Z Phenyl, gegebenenfalls substituiert mit 1 bis 3 Substituenten, die gleich oder verschieden sein können, ausgewählt aus der Gruppe, bestehend aus Hal, OH und C1-C4-Alkyl, darstellt; jedes R4 unabhängig Wasserstoff oder C1-C4-Alkyl darstellt, und n eine ganze Zahl von 1 bis 8 ist;
R1 ausgewählt ist aus der Gruppe, bestehend aus Cyclopentyl und Isopropyl, und die pharmazeutisch verträglichen Salze, optischen Isomeren und Hydrate davon. - Ebenfalls in den Bereich der Formel (I) fallen neuartige Verbindungen der Formel (Ic). worin R R2 darstellt,
R2 darstellt, worin Z einen Heterocyclus wie vorstehend definiert, gegebenenfalls substituiert mit 1 bis 3 Substituenten, die gleich oder verschieden sein können, ausgewählt aus der Gruppe, bestehend aus Hal, OH und C1-C4-Alkyl, darstellt; jedes R4 unabhängig Wasserstoff oder C1-C4-Alkyl darstellt, und n eine ganze Zahl von 1 bis 8 ist;
R1 ausgewählt ist aus der Gruppe, bestehend aus Cyclopentyl und Isopropyl, und die pharmazeutisch verträglichen Salze, optischen Isomeren und Hydrate davon. - Auch in den Bereich der Formel (I) fallen neuartige Verbindungen der Formel (Id), worin R R2 darstellt,
R2 darstellt, worin Z Cycloalkyl, gegebenenfalls substituiert mit 1 bis 3 Substituenten, die gleich oder verschieden sein können, ausgewählt aus der Gruppe, bestehend aus Hal, OH und C1-C4-Alkyl, darstellt; jedes R4 unabhängig Wasserstoff oder C1-C4-Alkyl darstellt, und n eine ganze Zahl von 1 bis 8 ist;
R1 ausgewählt ist aus der Gruppe, bestehend aus Cyclopentyl und Isopropyl, und die pharmazeutisch verträglichen Salze, optischen Isomeren und Hydrate davon. - Ebenfalls in den Bereich der Formel (I) fallen neuartige Verbindungen der Formel (Ia'). worin R R2NH- darstellt, worin
R2 C1-C8-Alkyl, gegebenenfalls substituiert mit 1 bis 3 Substituenten, die gleich oder verschieden sein können, die ausgewählt sind aus der Gruppe, bestehend aus Hal, OH, und C1-C4-Alkyl, darstellt;
R1 ausgewählt ist aus der Gruppe, bestehend aus Cyclopentyl und Isopropyl, und die pharmazeutisch verträglichen Salze, optischen Isomeren und Hydrate davon. - Auch in den Bereich der Formel (I) fallen neuartige Verbindungen der Formel (Ib'), worin R R2NH- darstellt,
R2 darstellt, worin Z Phenyl, gegebenenfalls substituiert mit 1 bis 3 Substituenten, die gleich oder verschieden sein können, ausgewählt aus der Gruppe, bestehend aus Hal, OH und C1-C4-Alkyl, darstellt; jedes R4 unabhängig Wasserstoff oder C1-C4-Alkyl darstellt, und n eine ganze Zahl von 1 bis 8 ist;
R1 ausgewählt ist aus der Gruppe, bestehend aus Cyclopentyl und Isopropyl, und die pharmazeutisch verträglichen Salze, optischen Isomeren und Hydrate davon. - Ebenfalls in den Bereich der Formel (I) fallen neuartige Verbindungen der Formel (Ic'). worin R R2NH- darstellt,
R2 darstellt, worin Z einen Heterocyclus wie vorstehend definiert, gegebenenfalls substituiert mit 1 bis 3 Substituenten, die gleich oder verschieden sein können, ausgewählt aus der Gruppe, bestehend aus Hal, OH und C1-C4-Alkyl, darstellt; jedes R4 unabhängig Wasserstoff oder C1-C4-Alkyl darstellt, und n eine ganze Zahl von 1 bis 8 ist;
R1 ausgewählt ist aus der Gruppe, bestehend aus Cyclopentyl und Isopropyl, und die pharmazeutisch verträglichen Salze, optischen Isomeren und Hydrate davon. - Ebenfalls in den Bereich der Formel (I) fallen neuartige Verbindungen der Formel (Id'). worin R R2NH- darstellt,
R2 darstellt, worin Z Cycloalkyl, gegebenenfalls substituiert mit 1 bis 3 Substituenten, die gleich oder verschieden sein können, ausgewählt aus der Gruppe, bestehend aus Hal, OH und C1-C4-Alkyl, darstellt; jedes R4 unabhängig Wasserstoff oder C1-C4-Alkyl darstellt, und n eine ganze Zahl von 1 bis 8 ist;
R1 ausgewählt ist aus der Gruppe, bestehend aus Cyclopentyl und Isopropyl, und die pharmazeutisch verträglichen Salze, optischen Isomeren und Hydrate davon. -
- Der hier verwendete Begriff „Heterocyclus" bedeutet eine beliebige geschlossene Ring-Einheit, in der ein oder mehrere der Atome des Rings ein anderes Element als Kohlenstoff sind und sie schließt die nachfolgenden ein, wobei sie nicht auf diese beschränkt ist: Piperidinyl, Pyridinyl, Isoxalyl, Tetrahydrofuranyl, Pyrrolidinyl, Morpholinyl, Piperazinyl; Benzimidazolyl; Thiazolyl; Thiophenyl; Furanyl; Indolyl, 1,3-Benzdioxolyl, Tetrahydropropanyl, Imidazolyl, Tetrahydrothiophenyl, Pyranyl, Dioxanyl, Pyrrolyl, Pyrimidinyl, Pyrazinyl, Triazinyl, Oxazolyl, Purinyl, Chinolinyl, und Isochinolinyl.
- Der hier verwendete Begriff „C1-C4-Alkyl" bezeichnet einen gesättigten oder ungesättigten, geradkettigen oder verzweigten Kohlenwasserstoff-Rest aus einem bis vier Kohlenstoffatomen und schließt die nachfolgenden ein, wobei er nicht auf diese beschränkt ist: Methyl, Ethyl, Propyl, Iso-propyl, 1-Propenyl, 2-Propenyl, n-Butyl, Isobutyl, tert-Butyl, sec-Butyl, 1-Butenyl, 2-Butenyl, 3-Butenyl, und ähnliche.
- Der hier verwendete Begriff „C1-C8-Alkyl" bezeichnet einen gesättigten oder ungesättigten, geradkettigen oder verzweigten Kohlenwasserstoff-Rest aus einem bis acht Kohlenstoffatomen und schließt die nachfolgenden ein, wobei er nicht auf diese beschränkt ist: Methyl, Ethyl, Propyl, Iso-propyl, 1-Propenyl, 2-Propenyl, n-Butyl, Isobutyl, tert-Butyl, sec-Butyl, 1-Butenyl, 2-Butenyl, 3-Butenyl, Pentyl, Neopentyl, Hexyl, Heptyl, Octyl und ähnliche.
- Der hier verwendete Begriff „C1-C8-Alkylen" bezeichnet einen gesättigten oder ungesättigten, geradkettigen oder verzweigten Kohlenwasserstoff-Rest aus einem bis acht Kohlenstoffatomen und schließt die nachfolgenden ein, wobei er nicht auf diese beschränkt ist: Methylen, Ethylen, Propylen, Isopropylen, 1-Propylen, 2-Propylen, n-Butylen, Isobutylen, tert-Butylen, sec-Butylen, 1-Butenylen, 2-Butenylen, 3-Butenylen, Pentylen, Neopentylen, Hexylen, Heptylen, Octylen und ähnliche.
- Der hier verwendete Begriff „Cycloalkyl" bezeichnet eine gesättigte oder ungesättigte alicyclische Einheit, enthaltend drei bis acht Kohlenstoffatome und schließt die nachfolgenden ein, wobei sie nicht auf diese beschränkt ist: Cyclopropyl, Cyclobutyl, Cyclopentyl, Cyclohexyl, Cycloheptyl, Cyclooctyl, und ähnliche.
- Der hier verwendete Begriff „Hal" bezeichnet eine Halogen-Einheit und schließt Fluor-, Chlor-, Brom- und Jod-Einheiten ein.
- Der hier verwendete Begriff „optisches Isomer" oder „optische Isomere" bezeichnet alle beliebigen der verschiedenen stereoisomeren Konfigurationen, die für eine gegebene Verbindung der Formel (I) existieren können.
- Der hier verwendete Begriff „Hydrat" oder „Hydrate" bezeichnet das Reaktionsprodukt aus einem oder mehreren Molekülen Wasser mit einer Verbindung der Formel (I), in der die H-OH-Bindung nicht aufgespalten ist und sie schließt Monohydrate als auch Multihydrate ein.
- Der hier verwendete Begriff „pharmazeutisch verträgliches Salz" bedeutet das Reaktionsprodukt aus einem oder mehreren Molekülen jeder beliebigen nicht-toxischen, organischen oder anorganischen Säure mit den Verbindungen der Formel (I). Veranschaulichungen für anorganische Säuren, die geeignete Salze bilden, schließen Salzsäure, Bromwasserstoffsäure, Schwefelsäure und Phosphorsäure und derartige Säuremetallsalze wie Natrium-monohydrogen-orthophosphat und Kaliumhydrogensulfat, ein. Veranschaulichungen für organische Säure, die geeignete Salze bilden, schließen Mono-, Di- und Tricarbonsäuren ein. Veranschaulichungen für derartige Säuren sind beispielsweise Essigsäure, Glykolsäure, Milchsäure, Brenztraubensäure, Malonsäure, Bernsteinsäure, Glutarsäure, Fumarsäure, Äpfelsäure, Weinsäure, Zitronensäure, Ascorbinsäure, Maleinsäure, Hydromaleinsäure, Benzoesäure, Hydrobenzoesäure, Phenylessigsäure, Zimtsäure, Salicylsäure, 2-Phenoxybenzoesäure und Sulfonsäuren, wie Methansulfonsäure, Trifluormethansulfonsäure und 2-Hydroxyethansulfonsäure, ein.
- Die Verbindungen der Formel (I) können unter Anwendung gut bekannter Verfahren und Techniken hergestellt werden und sind dem Fachmann bekannt. Ein allgemeines Syntheseschema zur Herstellung dieser Verbindungen wird in Schema A aufgeführt, wobei alle Substituenten, wenn nicht anders bezeichnet, wie vorstehend definiert sind.
- In Schema A, Schritt a, wird 2,6-Dichlorpurin (1) mit einem geeigneten Alkohol der Struktur 2 unter Entstehung der entsprechenden 9-substituierten 2,6-Dichlorpurin-Verbindung der Struktur 3 umgesetzt, unter Anwendung von Techniken und Verfahren, die dem Fachmann gut bekannt sind.
- Beispielsweise kann 2,6-Dichlorpurin (1) mit einem geeigneten Alkohol der Struktur 2 in Gegenwart von Triphenylphosphin und Diethylazodicarboxylat in einem geeigneten wasserfreien aprotischen Lösungsmittel, wie Tetrahydrofuran, umgesetzt werden. Die Reaktanten werden typischerweise gemeinsam bei Raumtemperatur über einen Zeitraum von 5 Stunden bis 5 Tagen gerührt. Das entstehende 9-substituierte 2,6-Dichlorpurin der Struktur 3 kann aus der Reaktionszone durch Extraktionsverfahren, die auf dem Fachgebiet bekannt sind gewonnen werden oder typischer wird das 9-substituierte 2,6-Dichlorpurin der Struktur 3 durch Entfernen des Lösungsmittel gewonnen, dem das direkte Auftragen auf eine Kieselgel-Säule und die Elution mit einem geeigneten Lösungsmittel, wie Methylenchlorid, oder einem Gemisch aus Lösungsmittel, wie einem Gemisch aus Hexan und Essigsäureethylester folgt. Das ungereinigte 9-substituierte 2,6-Dichlorpurin der Struktur 3 kann weiter durch Chromatografie aufgereinigt werden oder es kann in dem nächsten Schritt ohne weitere Aufreinigung verwendet werden.
- Im Schritt b wird die 6-Chlor-Funktionalität mit dem 9-substituierten 2,6-Dichlorpurin der Struktur 3 mit einem geeigneten Amin der Struktur 4 umgesetzt, was die entsprechende 9-substituierte 6-Amino-chlorpurin-Verbindung der Struktur 5 liefert.
- Beispielsweise kann das 9-substituierte 2,6-Dichlorpurin der Struktur 3 mit einem geeigneten Amin der Struktur 4 in einem geeigneten wasserfreien polaren Lösungsmittel, wie Methanol, umgesetzt werden. Die Reaktanten werden typischerweise gemeinsam bei Raumtemperatur über einen Zeitraum von 30 Minuten bis 3 Tagen unter Rückfluss gerührt. Das entstehende 9-substituierte 6-Amino-chlorpurin der Struktur 5 kann aus der Reaktionszone durch Extraktionsverfahren, die in dem Fachgebiet bekannt sind, gewonnen werden, oder wenn das 9-substituierte 6-Amino-chlorpurin der Struktur 5 aus der Lösung ausfällt, kann es durch Filtration gewonnen werden.
- Im Schritt c wird die 2-Chlor-Funktionalität des 9-substituierten 6-Amino-dichlorpurins der Struktur 5 mit 1,4-Cyclohexandiamin (6) umgesetzt, wobei die entsprechende Verbindung der Formel (I) entsteht.
- Beispielsweise kann ein geeignetes 9-substituiertes 6-Amino-chlorpurin der Struktur 5 mit einem molaren Überschuss an 1,4-Cyclohexandiamin (6) umgesetzt werden. Die Reaktanten werden typischerweise in ein Druckgefäß gegeben, verschlossen, und bei einer Temperatur von etwa 80°C bis etwa 150°C über einen Zeitraum von 30 Minuten bis 3 Tagen erhitzt. Die entstehende Verbindung der Formel (I) kann aus der Reaktionszone, durch Extraktionsverfahren, die auf dem Fachgebiet bekannt sind, gewonnen werden, und sie kann mittels Chromatografie aufgereinigt werden.
- Die in den allgemeinen, in Schema A aufgeführten Syntheseverfahren verwendeten Ausgangsmaterialien sind für den Fachmann leicht verfügbar.
- Die nachfolgenden Beispiele zeigen typische Synthesen, wie sind in Schema A beschrieben sind. Diese Beispiele sollten als Veranschaulichungen verstanden werden, und sie sollen den Bereich der vorliegenden Erfindung in keiner Weise einschränken. Die hier verwendeten nachfolgenden Begriffe besitzen die angezeigten Bedeutungen: „g" bedeutet Gramm; „mMol" bedeutet Millimol; „ml" bedeutet Milliliter; „Kp." bedeutet Siedepunkt; „°C" bedeutet Grad Celsius; „mmHg" bedeutet Millimeter an Quecksilber; „μl" bedeutet Mikroliter; „μg" bedeutet Mikrogramm; und „μM" bedeutet mikromolar.
- Beispiel 1
- 2-trans-[(4-Aminocyclohexyl)amino]-6-(3-jodbenzylamino)-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt a: 2,6-Dichlor-9-cyclopentylpurin
- Löse Cyclopentanol (260 mg, 3,02 mMol), 2,6-Dichlorpurin (680 mg, 3,60 mMol) und Triphenylphosphin (950 mg, 3,60 mMol) in trocknem THF (20 ml) und kühle auf 0°C. Setze Diethylazodicarboxylat (570 μl, 3,60 mMol) tropfenweise über einen Zeitraum von 15 Minuten unter einer Stickstoffatmosphäre zu. Rühre die entstehende Lösung 60 Stunden bei Raumtemperatur. Verdampfe das Lösungsmittel im Vakuum, lade es direkt auf eine Kieselgel-Säule, und eluiere mit Methylenchlorid unter Gewinnung der Titelverbindung als ungereinigtes Gemisch.
- Schema A, Schritt b: 2-Chlor-6-(3-jodbenzyl)amino)-9-cyclopentylpurine
- Löse 2,6-Dichlor-9-cyclopentylpurin (620 mg, Rohprodukt), 3-Jodbenzylaminhydrochlorid (810 mg, 3,00 mMol) und Triethylamin (835 μl, 6,00 mMol) in trocknem Ethanol (20 ml). Erhitze 15 Stunden unter Rückfluss, kühle ab, und filtriere den Feststoff unter Gewinnung der Titelverbindung als einen weißen Feststoff (680 mg).
'H-NMR (Me2SO-d6 + D2O, δ): 8,27 (s, 1H, Purin H-8), 7,74 (s, 1H), 7,61 (d, 1H), 7,39 (d, 1H), 7,16 (t, 1H), 4,78 (m, 1H), 4,62 (bs, 2H), 2,15 (m, 2H), 1,90 (m, 4H), 1,70 (m, 2H); CIMS (NH3) 454 (MH+), 328. - Schema A, Schritt c: [2-trans-(4-Aminocyclohexyl)amino]-6-(3-jodbenzylamino)-9-cyclopentylpurindihydrochlorid
- Mische 2-Chlor-6-(3-jodbenzyl)amino)-9-cyclopentylpurin (130 mg, 0,287 mMol) und 1,4-Cyclohexandiamin (2,00 g, Überschuss) in einem Druckgefäß, verschließe es und erhitze es 18 Stunden auf 140°C. Kühle das Reaktionsgemisch ab, setze CH2Cl2 (40 ml) zu, und wasche mit H2O (2 × 20 ml). Trockne es (MgSO4), dampfe das Lösungsmittel im Vakuum ab, und reinige durch Kieselgel-Chromatografie (10 : 1 : Tropfen CH2Cl2/MeOH/NH4OH), unter Gewinnung der Titelverbindung (140 mg, 92%). Wandle sie in das Hydrochloridsalz um.
1H-NMR (Me2SO-d6 + D2O, δ): 7,83 (d, 1H), 7,71 (s, 1H, Purin H-8), 7,60 (s, 1H), 7,38 (d, 1H), 7,14 (t, 1H), 4,63 (m, 3H), 3,62 (m, 1H), 2,99 (m, 1H), 1,50–2,20 (m, 14H), 1,10–1,50 (m, 2H);
CIMS (NH3) 532 (MH+). - Beispiel 2
- 2-trans-[(4-Aminocyclohexyl)amino]-6-[(3-indolyl)-2-ethylamino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(3-indolyl)-2-ethylamino]-9-cyclopentylpurin
- 2-Chlor-6-[(3-indolyl)-2-ethylamino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, Tryptamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-indolyl)-2-ethylamino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-indolyl)-2-ethylamino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(3-indolyl)-2-ethylamino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 459 (MH+); Rf (min.) = 3,47 - Beispiel 3
- 2-trans-[(4-Aminocyclohexyl)amino]-6-(butylamino)-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-(butylamino)-9-cyclopentylpurin
- 2-Chlor-6-(butylamino)-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, n-Butylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A. Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-(butylamino)-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-(butylamino)-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-(butylamino)-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 466 (MH+); Rf (min.) = 3,45 - Beispiel 4
- 2-[trans-(4-Aminocyclohexyl)amino-6-[2-(3,4-methylendioxyphenyl)ethylamino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[2-(3,4-methylendioxyphenyl)ethylamino]-9-cyclopentylpurin
- 2-Chlor-6-[2-(3,4-methylendioxyphenyl)ethylamino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 3,4-Methylendioxyphenethylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[2-(3,4-methylendioxyphenyl)ethylamino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[2-(3,4-methylendioxyphenyl)ethylamino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[2-(3,4-methylendioxyphenyl)ethylamino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 464 (MH+); Rf (min.) = 2,28 - Beispiel 5
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-aminobutyl)amino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(4-aminobutyl)amino]-9-cyclopentylpurin
- 2-Chlor-6-[(4-aminobutyl)amino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 1,4-Diaminobutan und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-[4-Aminocyclohexyl)amino]-6-[(4-aminobutyl)amino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-aminobutyl)amino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(4-aminobutyl)amino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 387 (MH+); Rf (min.) = 3,10 - Beispiel 6
- (S)-2-[trans-(4-Aminocyclohexyl)amino]-6-[(α-methylbenzyl)amino]-9-cyclopentpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(α-methylbenzyl)amino]-9-cyclopentylpurin
- 2-Chlor-6-[(α-methylbenzyl)amino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, (S)-α-Methylbenzylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(α-methylbenzyl)amino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(α-methylbenzyl)amino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(α-methylbenzyl)amino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 420 (MH+); Rf (min.) = 0,42 - Beispiel 7
- 2-[trans-[(4-Aminocyclohexyl)amino]-6-[(3-pyridyl)-2-ethylamino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(3-pyridyl)-2-ethylamino]-9-cyclopentylpurin
- 2-Chlor-6-[(3-pyridyl)-2-ethylamino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 3-(Ethylamino)pyridin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-pyridyl)-2-ethylamino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-pyridyl)-2-ethylamino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(3-pyridyl)-2-ethylamino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt
CIMS (NH3) 421 (MH+); Rf (min.) = 3,13 - Beispiel 8
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-pyridyl)methylamino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(4-pyridyl)methylamino]-9-cyclopentylpurin
- 2-Chlor-6-[(4-pyridyl)methylamino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 4-Aminomethylpyridin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-pyridyl)methylamino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-pyridyl)methylamino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor- 6-[(4-pyridyl)methylamino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 407 (MH+); Rf (min.) = 3,13 - Beispiel 9
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[3-(4-morpholinyl)propylamino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[3-(4-morpholinyl)propylamino]-9-cyclopentylpurin
- 2-Chlor-6-[3-(4-morpholinyl)propylamino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 3-Aminopropylmorpholin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[3-(4-morpholinyl)propylamino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[3-(4-morpholinyl)propylamino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[3-(4-morpholinyl)propylamino]-9-cyclopentylpurin wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 443 (MH+); Rf (min.) = 3,11 - Beispiel 10
- 2-[trans-[(4-Aminocyclohexyl)amino]-6-[(3,4-dichlorbenzyl)amino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(3,4-dichlorbenzyl)amino]-9-cyclopentylpurin
- 2-Chlor-6-[(3,4-dichlorbenzyl)amino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 3,4-Dichlorbenzylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3,4-dichlorbenzyl)amino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3,4-dichlorbenzyl)amino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(3,4-dichlorbenzyl)amino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 474 (MH+); Rf (min.) = 2,34 - Beispiel 11
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-methylbenzyl)amino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(3-methylbenzyl)amino]-9-cyclopentylpurin
- 2-Chlor-6-[(3-methylbenzyl)amino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 3-Methylbenzylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-methylbenzyl)amino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-methylbenzyl)amino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(3-methylbenzyl)amino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 420 (MH+); Rf (min.) = 2,29 - Beispiel 12
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[2-(2-pyridyl)ethylamino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[2-(2-pyridyl)ethylamino]-9-cyclopentylpurin
- 2-Chlor-6-[2-(2-pyridyl)ethylamino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 2-(Ethylamino)pyridin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)-amino]-6-[2-(2-pyridyl)ethylamino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[2-(2-pyridyl)ethylamino]-9-cyclopentylpurinhydrochlorid wird aus 2-Chlor-6-[2-(2-pyridyl)ethylamino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 421 (MH+); Rf (min.) = 3,13 - Beispiel 13
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[2-(4-morpholinyl)ethylamino]-9-cyclopentylpurintrihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[2-(4-morpholinyl)ethylamino]-9-cyclopentylpurin
- 2-Chlor-6-[2-(4-morpholinyl)ethylamino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 2-Aminoetylmorpholin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[2-(4-morpholinyl)ethylamino]-9-cyclopentylpurintrihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[2-(4-morpholinyl)ethylamino]-9-cyclopentylpurintrihydrochlorid wird aus 2-Chlor-6-[2-(4-morpholinyl)ethylamino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 429 (MH+); Rf (min.) = 3,08 - Beispiel 14
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[2-hydroxyethylhydrazino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[2-hydroxyethylhydrazino]-9-cyclopentylpurin
- 2-Chlor-6-[2-hydroxyethylhydrazino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 2-Hydroxyethylhydrazin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A. Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[2-hydroxyethylhydrazino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[2-hydroxyethylhydrazino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[2-hydroxyethylhydrazino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 375 (MH+); Rf (min.) = 3,15 - Beispiel 15
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-chlor)benzylamino]-9-(2-propyl)purindihydrochlorid
- Schema A, Schritt a: 2,6-Dichlor-9-(2-propyl)purin
- 2,6-Dichlor-9-(2-propyl)purin wird aus 2,6-Dichlorpurin und Isopropanol im Wesentlichen wie in Beispiel 1, Schema A, Schritt a beschrieben hergestellt, wobei das Cyclopentanol jedoch durch Isopropanol ersetzt wird.
- Schema A, Schritt b: 2-Chlor-6-[(3-chlor)benzylamino]-9-(2-propyl)purin
- 2-Chlor-6-[(3-chlor)benzylamino]-9-(2-propyl)purin wird aus 2,6-Dichlor-9-(2-propyl)purin, 3-Chlorbenzylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A. Schritt c: 2-[trans-(4-Aminocyclohexyl)-amino]-6-[(3-chlor)benzylamino]-9-(2-propyl)purindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-chlor)benzylamino]-9-(2-propyl)purindihydrochlorid wird aus 2-Chlor-6-[(3-chlor)benzylamino]-9-(2-propyl)purin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 414 (MH+); Rf (min.) = 3,44 - Beispiel 16
- (R)-2-[trans-(4-Aminocyclohexyl)amino]-6-[(α-methylbenzyl)amino]-9-cyclopentylpurindihydrochlorid
- Schema A. Schritt b: 2-Chlor-6-[(α-methylbenzyl)amino]-9-cyclopentylpurin
- 2-Chlor-6-[(α-methylbenzyl)amino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, (R)-α-Methylbenzylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(α-methylbenzyl)amino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(α-methylbenzyl)amino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(α-methylbenzyl)amino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 420 (MH+); Rf (min.) = 2,27 - Beispiel 17
- 2-[trans-(4-Aminocyclohexyl)amino]-6-(2-thiophenmethylamino)-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-(2-thiophenmethylamino)-9-cyclopentylpurin
- 2-Chlor-6-(2-thiophenmethylamino)-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 2-Thiophenmethylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)-amino]-6-(2-thiophenmethylamino)-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-(2-thiophenmethylamino)-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-(2-thiophenmethylamino)-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 412 (MH+); Rf (min.) = 3,43 - Beispiel 18
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(2-chlorbenzyl)amino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(2-chlorbenzyl)amino]-9-cyclopentylpurin
- 2-Chlor-6-[(2-chlorbenzyl)amino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 2-Chlorbenzylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(2-chlorbenzyl)amino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(2-chlorbenzyl)amino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(2-chlorbenzyl)amino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 440 (MH+); Rf (min.) = 2,28 - Beispiel 19
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(2-benzimidazolyl)methylamino]-9-cyclopentylpurindihydrochlorid
- Schema A. Schritt b: 2-Chlor-6-[(2-benzimidazolyl)methylamino]-9-cyclopentylpurin
- 2-Chlor-6-[(2-benzimidazolyl)methylamino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 2-(Methylamino)benzimidazol und Triethylamin im Wesentlichen wie vor stehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A. Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(2-benzimidazolyl)methylamino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(2-benzimidazolyl)methylamino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(2-benzimidazolyl)methylamino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 372 (MH+); Rf (min.) = 3,4 - Beispiel 20
- 2-[trans-(4-Aminocyclohexyl)amino]-6-(octylamino)-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-(octylamino)-9-cyclopentylpurin
- 2-Chlor-6-(octylamino)-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, n-Octylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-(octylamino)-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-(octylamino)-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-(octylamino)-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 428 (MH+); Rf (min.) = 4,23 - Beispiel 21
- 2-[trans-(4-Aminocyclohexyl)amino]-6-(4-phenylbutylamino)-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-(4-phenylbutylamino)-9-cyclopentylpurin
- 2-Chlor-6-(4-phenylbutylamino)-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 4-Phenylbutylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A. Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-(4-phenylbutylamino)-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-(4-phenylbutylamino)-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-(4-phenylbutylamino)-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 448 (MH+); Rf (min.) = 4,09 - Beispiel 22
- 2-[trans-(4-Aminocycohexyl)amino]-6-[(cyclohexyl)methylamino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(cyclohexyl)methylamino]-9-cyclopentylpurin
- 2-Chlor-6-[(cyclohexyl)methylamino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, Aminomethylcyclohexan und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(cyclohexyl)methylamino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(cyclohexyl)methylamino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(cyclohexyl)methylamino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 412 (MH+); Rf (min.) = 2,33 - Beispiel 23
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-(3-methyl-4-hydroxy)butyl)amino]-9-cyclopentylpurindihydrochlorid
- Schema A. Schritt b: 2-Chlor-6-[(3-(3-methyl-4-hydroxy)butyl)amino]-9-cyclopentylpurin
- 2-Chlor-6-[(3-(3-methyl-4-hydroxy)butyl)amino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 2-(2-Hydroxymethyl)butylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A. Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-(3-methyl-4-hydroxy)butyl)amino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-(3-methyl-4-hydroxy)butyl)amino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(3-(3-methyl-4-hydroxy)butyl)amino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 404 (MH+); Rf (min.) = 3,15 - Beispiel 24
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[3-(phenyl)propylamino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[3-(phenyl)propylamino]-9-cyclopentylpurin
- 2-Chlor-6-[3-(phenyl)propylamino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 3-Aminopropylbenzol und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[3-(phenyl)propylamino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[3-(phenyl)propylamino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[3- (phenyl)propylamino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 434 (MH+); Rf (min.) = 0,49 - Beispiel 25
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[5-(hydroxy)pentylamino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[5-(hydroxy)pentylamino]-9-cyclopentylpurin
- 2-Chlor-6-[5-(hydroxy)pentylamino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 5-Hydroxypentylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[5-(hydroxy)pentylamino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[5-(hydroxy)pentylamino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[5-(hydroxy)pentylamino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 402 (MH+); Rf (min.) = 3,25 - Beispiel 26
- 2-[trans-(4-Aminocyclohexyl)amino]-6-(pentylamino)-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-(pentylamino)-9-cyclopentylpurin
- 2-Chlor-6-(pentylamino)-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, Pentylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-(pentylamino)-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-(pentylamino)-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-(pentylamino)-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 386 (MH+); Rf (min.) = 3,52 - Beispiel 27
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-chlorbenzyl)amino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(4-chlorbenzyl)amino]-9-cyclopentylpurin
- 2-Chlor-6-[(4-chlorbenzyl)amino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 4-Chlorbenzylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-chlorbenzyl)amino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-chlorbenzyl)amino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(4-chlorbenzyl)amino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 440 (MH+); Rf (min.) = 2,29 - Beispiel 28
- 2-[trans-(4-Aminocyclohexyl)amino]-6-(methylamino)-9-cylopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-(methylamino)-9-cyclopentylpurin
- 2-Chlor-6-(methylamino)-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, Methylaminhydrochlorid und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)-amino]-6-(methylamino)-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-(methylamino)-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-(methylamino)-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 330 (MH+); Rf (min.) = 3,15 - Beispiel 29
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-chlorbenzyl)amino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(3-chlorbenzyl)amino]-9-cyclopentylpurin
- 2-Chlor-6-[(3-chlorbenzyl)amino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 3-Chlorbenzylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-chlorbenzyl)amino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-chlorbenzyl)amino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(3-chlorbenzyl)amino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 440 (MH+); Rf (min.) = 2,30 - Beispiel 30
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(2-tetrahydropyranyl)methylamino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(2-tetrahydropyranyl)methylamino]-9-cyclopentylpurin
- 2-Chlor-6-[(2-tetrahydropyranyl)methylamino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 2-Aminomethyltetrahydropyran und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(2-tetrahydropyranyl)methylamino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(2-tetrahydropyranyl)methylamino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(2-tetrahydropyranyl)methylamino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 414 (MH+); Rf (min.) = 3,39 - Beispiel 31
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-pyridyl)-2-ethylamino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(4-pyridyl)-2-ethylamino]-9-cyclopentylpurin
- 2-Chlor-6-[(4-pyridyl)-2-ethylamino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 4-(Ethylamino)pyridin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)-amino]-6-[(4-pyridyl)-2-ethylamino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-pyridyl)-2-ethylamino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(4-pyridyl)-2-ethylamino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 421 (MH+); Rf (min.) = 3,13 - Beispiel 32
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(cyclopropyl)methylamino]-9-(2-propyl)purindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(cyclopropyl)methylamino]-9-(2-propyl)purin
- 2-Chlor-6-[(cyclopropyl)methylamino]-9-(2-propyl)purin wird aus 2,6-Dichlor-9-(2-propyl)purin (für die Herstellung siehe Beispiel 15), Cyclopropylmethylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(cyclopropyl)methylamino]-9-(2-propyl)purindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(cyclopropyl)methylamino]-9-(2-propyl)purindihydrochlorid wird aus 2-Chlor-6-[(cyclopropyl)methylamino]-9-(2-propyl)purin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 344 (MH+); Rf (min.) = 3,25 - Beispiel 33
- 2-[trans-(4-Aminocyclohexyl)amino]-6-(ethylamino)-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-(ethylamino)-9-cyclopentylpurin
- 2-Chlor-6-(ethylamino)-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, Ethylaminhydrochlorid und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)-amino]-6-(ethylamino)-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-(ethylamino)-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-(ethylamino)-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 472 (MH+); (min.) = 3,46 - Beispiel 34
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(cyclopropyl)methylamino]-9-cyclopentpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(cyclopropyl)methylamino]-9-cyclopentylpurin
- 2-Chlor-6-[(cyclopropyl)methylamino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, Aminomethylcyclopropan und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(cyclopropyl)methylamino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(cyclopropyl)methylamino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(cyclopropyl)methylamino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 370 (MH+); Rf (min.) = 2,21 - Beispiel 35
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[2-phenethylhydrazino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[2-phenethylhydrazino]-9-cyclopentylpurin
- 2-Chlor-6-[2-phenethylhydrazino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 2-Phenylethylhydrazin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[2-phenethylhydrazino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[2-phenethylhydrazino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[2- phenethylhydrazino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 435 (MH+); (min.) = 3,54 - Beispiel 36
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-methylbutyl)amino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(3-methylbutyl)amino]-9-cyclopentylpurin
- 2-Chlor-6-[(3-methylbutyl)amino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 3-Methylbutylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-[(4-Aminocyclohexyl)amino]-6-[(3-methylbutyl)amino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-methylbutyl)amino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(3-methylbutyl)amino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 386 (MH+); Rf (min.) = 3,53 - Beispiel 37
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(butyl)amino]-9-(2-propyl)purindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(butyl)amino]-9-(2-propyl)purin
- 2-Chlor-6-[(butyl)amino]-9-(2-propyl)purin wird aus 2,6-Dichlor-9-(2-propyl)purin (für die Herstellung siehe Beispiel 15), Butylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(butyl)amino]-9-(2-propyl)purindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(butyl)amino]-9-(2-propyl)purindihydrochlorid wird aus 2-Chlor-6-[(butyl)amino]-9-(2-propyl)purin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 346 (MH+); (min.) = 3,33 - Beispiel 38
- 2-[trans-(4-Aminocyclohexyl)amino]-6-(2-furanmethylamino)-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-(2-furanmethylamino)-9-cyclopentylpurin
- 2-Chlor-6-(2-furanmethylamino)-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, Furfurylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A. Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-(2-furanmethylamino)-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-(2-furanmethylamino)-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-(2-furanmethylamino)-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 396 (MH+); (min.) = 3,38 - Beispiel 39
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-(2-imidazoyl)propyl)amino]-9-(2-propyl)purintrihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(3-(2-imidazoyl)propyl)amino]-9-(2-propyl)purin
- 2-Chlor-6-[(3-(2-imidazoyl)propyl)amino]-9-(2-propyl)purin wird aus 2,6-Dichlor-9-(2-propyl)purin (für die Herstellung siehe Beispiel 19), 3-(2-Imidazolyl)propylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A. Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-(2-imidazoyl)propyl)amino]-9-(2-propyl)purintrihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-(2-imidazoyl)propyl)amino]-9-(2-propyl)purintrihydrochlorid wird aus 2-Chlor-6-[(3-(2-imidazoyl)propyl)amino]-9-(2-propyl)purin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 398 (MH+); Rf (min.) = 3,01 - Beispiel 40
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-chlor-2-fluorbenzyl)amino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(4-chlor-2-fluorbenzyl)amino]-9-cyclopentylpurin
- 2-Chlor-6-[(4-chlor-2-fluorbenzyl)amino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 4-Chlor-2-fluorbenzylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-chlor-2-fluorbenzyl)amino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-chlor-2-fluorbenzyl)amino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(4-chlor-2-fluorbenzyl)amino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 458 (MH+); Rf (min.) = 2,31 - Beispiel 41
- 2-[trans-(4-Aminocyclohexyl)amino]-6-(hexylamino)-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-(hexylamino)-9-cyclopentylpurin
- 2-Chlor-6-(hexylamino)-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, n-Hexylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-(hexylamino)-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-(hexylamino)-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-(hexylamino)-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 400 (MH+); Rf (min.) = 4,03 - Beispiel 42
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(2-fluorbenzyl)amino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(2-fluorbenzyl)amino]-9-cyclopentylpurin
- 2-Chlor-6-[(2-fluorbenzyl)amino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 2-Fluorbenzylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[2-fluorbenzyl)amino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(2-fluorbenzyl)amino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(2-fluorbenzyl)amino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 424 (MH+); Rf (min.) = 2,22 - Beispiel 43
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[2-(phenyl)ethylamino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[2-(phenyl)ethylamino]-9-cyclopentylpurin
- 2-Chlor-6-[2-(phenyl)ethylamino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 2-Aminoethylbenzol und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[2-(phenyl)ethylamino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[2-(phenyl)ethylamino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[2-(phenyl)ethylamino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 420 (MH+); Rf (min.) = 0,43 - Beispiel 44
- 2-[trans-(4-Aminocyclohexyl)amino]-6-(propylamino)-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-(propylamino)-9-cyclopentylpurin
- 2-Chlor-6-(propylamino)-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, n-Propylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-(propylamino)-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-(propylamino)-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-(propylamino)-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 358 (MH+); Rf (min.) = 3,31 - Beispiel 45
- 2-[trans-(4-Aminocyclohexyl)amino]-6-(benzylamino)-9-(2-propyl)purintrihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-(benzylamino)-9-(2-propyl)purin
- 2-Chlor-6-(benzylamino)-9-(2-propyl)purin wird aus 2,6-Dichlor-9-(2-propyl)purin (für die Herstellung siehe Beispiel 15), Benzylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-(benzylamino)-9-(2-propyl)purindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-(benzylamino)-9-(2-propyl)purindihydrochlorid wird aus 2-Chlor-6-(benzylamino)-9-(2-propyl)purin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 380 (MH+); Rf (min.) = 3,34 - Beispiel 46
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[3-(1-imidazolyl)propylamino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[3-(1-imidazolyl)propylamino]-9-cyclopentylpurin
- 2-Chlor-6-[3-(1-imidazolyl)propylamino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 1-(3-Aminopropyl)imidazol und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[3-(1-imidazolyl)propylamino]-9-cyclopentylpurindihdrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[3-(1-imidazolyl)propylamino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[3-(1-imidazolyl)propylamino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 424 (MH+); Rf (min.) = 3,13 - Beispiel 47
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[benzylamino]-9-clopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[benzylamino]-9-cyclopentylpurin
- 2-Chlor-6-[benzylamino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, Benzylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[benzylamino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[benzylamino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[benzylamino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 406 (MH+); Rf (min.) = 2,23 - Beispiel 48
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(2,4-dichlorbenzyl)amino]-9-cyclopentylpurindihydrochlorid
- Schema A. Schritt b: 2-Chlor-6-[(2,4-dichlorbenzyl)amino]-9-cyclopentylpurin
- 2-Chlor-6-[(2,4-dichlorbenzyl)amino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 2,4-Dichlorbenzylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A. Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(2,4-dichlorbenzyl)amino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(2,4-dichlorbenzyl)amino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor- 6-[(2,4-dichlorbenzyl)amino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 474 (MH+); Rf (min.) = 2,34 - Beispiel 49
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[2,2,2-trifluorethylamino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[2,2,2-trifluorethylamino]-9-cyclopentylpurin
- 2-Chlor-6-[2,2,2-trifluorethylamino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 2,2,2-Trifluorethylaminhydrochlorid und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[2,2,2-trifluorethylamino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[2,2,2-trifluorethylamino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[2,2,2-trifluorethylamino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 398 (MH+); Rf (min.) = 3,33 - Beispiel 50
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-fluorbenzyl)amino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(4-fluorbenzyl)amino]-9-cyclopentylpurin
- 2-Chlor-6-[(4-fluorbenzyl)amino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 4-Fluorbenzylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-fluorbenzyl)amino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-fluorbenzyl)amino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(4-fluorbenzyl)amino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 424 (MH+); Rf (min.) = 2,24 - Beispiel 51
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-jodbenzyl)amino]-9-(2-propyl)purindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(3-jodbenzyl)amino]-9-(2-propyl)purin
- 2-Chlor-6-[(3-jodbenzyl)amino]-9-(2-propyl)purin wird aus 2,6-Dichlor-9-(2-propyl)purin (für die Herstellung siehe Beispiel 15), 3-Jodbenzylamin und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-jodbenzyl)amino]-9-(2-propyl)purindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-jodbenzyl)amino]-9-(2-propyl)purindihydrochlorid wird aus 2-Chlor-6-[(3-jodbenzyl)amino]-9-(2-propyl)purin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 506 (MH+); Rf (min.) = 3,53 - Beispiel 52
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[2,2,2-trifluorethylhydrazino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[2,2,2-trifluorethylhydrazino]-9-cyclopentylpurin
- 2-Chlor-6-[2,2,2-trifluorethylhydrazino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 2,2,2-Trifluorethylhydrazin und Triethylamin im Wesentlichen wie vor stehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[2,2,2-trifluorethylhydrazino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[2,2,2-trifluorethylhydrazino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[2,2,2-trifluorethylhydrazino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 413 (MH+); Rf (min.) = 3,28 - Beispiel 53
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-hydroxypropyl)amino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(3-hydroxypropyl)amino]-9-cyclopentylpurin
- 2-Chlor-6-[(3-hydroxypropyl)amino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 3-Amino-1-propanol und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-hydroxypropyl)amino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-hydroxypropyl)amino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(3-hydroxypropyl)amino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 374 (MH+); Rf (min.) = 3,17 - Beispiel 54
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(5-hydroxy-1,5-dimethylhexyl)amino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(5-hydroxy-1,5-dimethylhexyl)amino]-9-cyclopentylpurin
- 2-Chlor-6-[(5-hydroxy-1,5-dimethylhexyl)amino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 6-Amino-2-methyl-2-heptanolhydrochlorid und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(5-hydroxy-1,5-dimethylhexyl)amino]-9-clopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(5-hydroxy-1,5-dimethylhexyl)amino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(5-hydroxy-1,5-dimethylhexyl)amino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 444 (MH+); Rf (min.) = 3,37 - Beispiel 55
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-hydroxyphenyl)ethylamino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(4-hydroxyphenyl)ethylamino]-9-cyclopentylpurin
- 2-Chlor-6-[(4-hydroxyphenyl)ethylamino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, Tyraminhydrochlorid und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-hydroxyphenyl)ethylamino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-hydroxyphenyl)ethylamino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(4-hydroxyphenyl)ethylamino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben, hergestellt.
CIMS (NH3) 436 (MH+); Rf (min.) = 3,38 - Beispiel 56
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(8-aminooctyl)amino]-9-cyclopentylpurindihydrochlorid
- Schema A, Schritt b: 2-Chlor-6-[(8-aminooctyl)amino]-9-cyclopentylpurin
- 2-Chlor-6-[(8-aminooctyl)amino]-9-cyclopentylpurin wird aus 2,6-Dichlor-9-cyclopentylpurin, 1,8-Diaminooctan und Triethylamin im Wesentlichen wie vorstehend in Beispiel 1, Schema A, Schritt b beschrieben hergestellt.
- Schema A, Schritt c: 2-[trans-(4-Aminocyclohexyl)amino)-6-[(8-aminooctyl)amino]-9-cyclopentylpurindihydrochlorid
- 2-[trans-(4-Aminocyclohexyl)amino]-6-[(8-aminooctyl)amino]-9-cyclopentylpurindihydrochlorid wird aus 2-Chlor-6-[(8-aminooctyl)amino]-9-cyclopentylpurin im Wesentlichen wie in Beispiel 1, Schema A, Schritt c beschrieben hergestellt.
CIMS (NH3) 443 (MH+); Rf (min.) = 3,30 - Der hier verwendete Begriff "neoplastischer Krankheitszustand" bezeichnet einen abnormalen Zustand oder eine abnormale Bedingung, der/die durch unkontrollierte Proliferation charakterisiert ist. Neoplastische Krankheitszustände schließen Leukämien, Karzinome und Adenokarzinome, Sarkome, Melanome und neoplasmamische Typen ein.
- Leukämien schließen akute lymphoblastische, chronische lymphozytische, akute myeloblastische und chronische myelozytische Leukämien ein, sie sind jedoch nicht darauf beschränkt.
- Karzinome und Adenokarzinome schließen jene des Gebärmutterhalses, der Brust, der Prostata, des Ösophagus, des Magens, des Dünndarms, des Kolons, des Ovariums und der Lungen ein, sie sind jedoch nicht darauf beschränkt.
- Sarkome schließen Österome, Osteosarkom, Lipom, Lipsarkom, Hämangiome und Hämangiosarkom ein, sie sind jedoch nicht darauf beschränkt.
- Melanome schließen amelanotische und melanotische Melanome ein, sie sind jedoch nicht darauf beschränkt.
- Neoplasmamische Typen schließen Karzinosarkom, einen lymphoiden Gewebstyp, follikuläres Retikulum, Zellsarkom und Hodgkinsche Krankheit ein, sie sind jedoch nicht darauf beschränkt.
- Der Begriff "therapeutisch wirksame Menge" einer Verbindung der Formel (I) bezeichnet eine Menge, die nach einfacher oder mehrfacher Verabreichung einer Dosis an einen Patienten bei der Kontrolle des Wachstums des Neoplasmas oder von Metastasen des Neoplasmas oder zum Schutz vor Apoptose wirksam ist. Eine therapeutisch wirksame Menge einer Verbindung der Formel variiert entsprechend dem Alter, dem Gewicht, dem Typ des zu behandelnden Neoplasmas, der Kombination anderer antineoplastischer Mittel und entsprechend anderer Kriterien, die dem Fachmann unter Verwendung herkömmlicher klinischer und laborklinischer Verfahren gut bekannt sind. Eine therapeutisch wirksame Menge einer Verbindung der Formel variiert entsprechend dem für die Apoptose anfälligen Zelltyp, der Lokalisation des Infarkts als auch dem Alter, Gewicht und anderer dem Fachmann gut bekannter Kriterien.
- Der Begriff "Kontrollieren bzw. Bekämpfen des Wachstums" des Neoplasmas bezeichnet das Verlangsamen, das Unterbrechen, das in die Ruhelage versetzen oder das Beenden des Wachstums des Neoplasmas oder der Metastasen des Neoplasmas. Der Begriff "Kontrollieren des Wachstums" des Neoplasmas bezeichnet auch das Abtöten der Neoplasie oder der Metastasen der Neoplasie.
- Eine wirksame Menge einer Verbindung der Formel ist die Menge, die nach einfacher oder mehrfacher Verabreichung einer Dosis an einen Patienten bei der Bereitstellung einer antineoplastischen Wirkung oder zum Schutz vor Apoptose wirksam ist. Eine "antineoplastische Wirkung" bezeichnet das Verlangsamen, das Unterbrechen oder Zerstören des weiteren Wachstums von neoplastischen Zellen.
- Eine wirksame antineoplastische Menge einer Verbindung der Formel kann durch einen auf dem Fachgebiet ausgebildeten behandelnden Diagnostiker durch Anwendung bekannter Verfahren und indem die der unter ähnlichen Umständen erhaltenen Ergebnisse in Betracht gezogen werden, leicht ermittelt werden. Zur Bestimmung der wirksamen Menge wird durch den behandelnden Diagnostiker eine Vielzahl von Faktoren in Betracht gezogen, einschließlich des Typs des Säugetiers; seiner Größe, seines Alters und seines Allgemeinzustands; der spezifischen Erkrankung; des Schweregrads der Erkrankung; der Ansprechbarkeit des individuellen Patienten; der besonderen verabreichten Verbindung der Formel; des Modus der Verabreichung; der Charakteristika der Bioverfügbarkeit des verabreichten Präparats; des ausgewählten Dosierungsregimes; der Verwendung von begleitender Medikation; und anderer relevanter Begleitumständen, sie sind jedoch nicht auf diese beschränkt.
- Eine weitere Ausführungsform der vorliegenden Erfindung schließt ein Verfahren zur prophylaktischen Behandlung eines Patienten ein, der ein Risiko zur Entwicklung eines neoplastischen Krankheitszustands aufweist, umfassend die Verabreichung einer prophylaktisch wirksamen antineoplastischen Menge einer Verbindung der Formel. Der Begriff "Patient mit einem Risiko zur Entwicklung eines neoplastischen Krankheitszustands" bezeichnet einen Patienten, der infolge einer identifizierten genetischen Veranlagung für Neoplasmen, Neoplasmen aufwies, karzinogem Mittel ausgesetzt war, Ernährungsweise, oder anderer mit der Entwicklung von neoplastischen Krankheitszuständen assoziierter Risikofaktoren aufwies oder derzeit aufweist. Bevorzugte Patienten mit einem Risiko zur Entwicklung eines neoplastischen Krankheitszustands schließen Patienten ein, die bezüglich onkogener Viren postiv sind, sich nach einer früheren Behandlung wegen eines/von Neoplasma/Neoplasmen in Remis sion befinden, Tabakprodukte konsumieren oder früher Karzinogenen, wie Asbest, ausgesetzt waren, oder bezüglich anderer neoplastischer genetischer Marker postiv sind.
- Onkogene Viren sind jene Viren, die mit Krebs einhergehen. Beispielsweise wurde festgestellt, dass das Rous-Sarkom bei Hühnern, das Shope-Papillom bei Kaninchen, Leukämie-Viren bei Mäusen, tierische Viren sind, sie eine Rolle bei der Entwicklung verschiedener Krebsarten haben. Das humane Papillom-Virus geht mit Genitalkrebs einher. Das Molluscum contagiosum-Virus geht mit Molluscum contagiosum Tumoren einher. Das JC-Virus, ein humanes Papovirus, geht mit Erkrankungen des retikulendothelialen Systems, wie Leukämie und Lymphom, einher. Humane Retroviren, wie die humanen lymphotropen T-Zell-Viren (HTLV) der Typen 1 und 2 gehen mit einigen humanen Leukämien und Lymphomen einher. Die humanen immundefizitären Viren (HIV) der Typen 1 und 2 sind die Ursache von AIDS. Das Epstein-Barr-Virus geht mit verschiedenen bösartigen Erkrankungen einher, einschließlich demnasopharyngealen Karzinom, dem Afrikanischen Burkitt-Lymphom und Lymphomen bei Empfängern von Organtransplantaten, die mit Immunseppressiva behandelt werden.
- Genetische Marker, wie Mutationen, Umordnungen und ähnliche in BRCA1, bcl-1/PRAD1, Cyclin D1/CCND1, p16, cdk4, insbesondere eine Arg24Cys-Mutation, p16INK4a gehen mit Veranlagungen bezüglich verschiedener Neoplasmen einher. Beispielsweise gehen Veränderungen in dem BRCA1-Gen mit einem erhöhten Risiko für Brust- und Ovarienkrebs einher. Andere genetische Marker schließen Veränderungen in dem MMSC1-Gen, das mit dem MMCA1-Gehirn- und Prostata-Krebs-Gen in Wechselwirkung steht, in dem CtIP-Gen, das mit dem BRACA1-Gen bei Brust- und Ovarien-Krebs verbunden ist, und an das BRCA1-Gen bindet und das mit dem E1A-Onkogen-Pathway verbunden ist, und in dem MKK3-Gen, das ein Kontrollgen des Zellzyklus ist, das als Tumorsuppressor bei Lungenkrebs wirkt, indem es die Apoptose aktiviert, ein. Patienten mit einem Risiko für die Entwicklung eines neoplastischen Krankheitszustands schließen auch Patienten ein, die verschiedene Zellzyklus-Proteine überexprimieren, einschließlich cdk4 und die Cycline B1 und E. Patienten mit einem Risiko für die Entwicklung eines neoplastischen Krankheitszustands schließen jene mit erhöhten Spiegeln an Tumormarkern ein. Bekannte Tumormarker schließen Prostata-spezifisches Antigen (PSA) und Plasma Insulin-ähnliche Wachstumsfaktor-1 (IGF-1) ein, die Marker für Prostatakrebs sind. Nukleare Matrixproteine (NMPs) gehen mit der Anwesenheit von Krebs einher, insbeondere mit Blasen- und Kolonkrebs.
- Es wird erwartet, dass eine wirksame Menge einer Verbindung der Formel von etwa 25 Nanogramm pro Kilogramm Körpergewicht pro Tag (ng/kg/Tag) bis etwa 500 mg/kg/Tag variiert. Bevorzugte wirksame Mengen einer Verbindung der Formel betragen etwa 1 μg/kg/Tag bis etwa 500 μg/kg/Tag. Eine bevorzugtere Menge einer Verbindung der Formel beträgt etwa 1 μg/kg/Tag bis etwa 50 μg/kg/Tag.
- Eine Verbindung der Formel kann in jeder beliebigen Form und auf jedem beliebigen Weg verabreicht werden, der die Bioverfügbarkeit der Verbindung in wirksamen Mengen sicherstellt. Verbindungen der Formel können auf oralen oder parenteralen Wegen verabreicht werden. Verbindungen der Formel können oral, subkutan, intramuskulär, intravenös, transdermal, intranasal, rektal, okular und in ähnlicher Weise verabreicht werden. Die orale Verabreichung ist bevorzugt. Ein Fachmann auf dem Gebiet der Herstellung pharmazeutischer Formulierungen kann leicht die geeigneten Formen einer Verbindung der Formel ermitteln, indem die besonderen Charakteristika der Verbindung, die zu behandelnde Krankheit, der Krankheitszustand, die Ansprechbarkeit anderer Patienten oder andere relevante Begleitumstände ermittelt werden.
- Eine Verbindung der Formel kann mit Trägern, Exzipienten oder anderen Verbindungen zur Herstellung von Zusammensetzungen einer Verbindung der Formel kombiniert werden. Eine Verbindung der Formel kann eine Verbindung der Formel in Anmi schung oder anderweitig in Verbindung mit einem oder mehreren inerten Trägern umfassen. Zusammensetzungen der Formel sind beispielsweise als geeignete Mittel zum Transportieren loser Ware oder zur Lagerung einer Verbindung der Formel nützlich. Ein inerter Träger ist ein Material, das sich nicht zersetzt oder anderweitig kovalent mit einer Verbindung der Formel reagiert. Ein inerter Träger kann ein festes, ein halbfestes oder ein flüssiges Material sein. Bevorzugte Träger sind Wasser, wässrige Puffer, organische Lösungsmittel und pharmazeutisch verträgliche Träger oder Exzipienten. Bevorzugte wässrige Puffer stellen einen Pufferbereich bereit, in dem die Verbindung der Formel sich nicht zersetzt. Bevorzugte Pufferbereiche liegen zwischen etwa pH 4 bis etwa pH 9. Bevorzugte organische Lösungsmittel sind Acetonitril, Essigsäureethylester, Hexan.
- Eine pharmazeutische Zusammensetzung einer Verbindung der Formel umfasst eine Verbindung der Formel in Anmischung oder anderweitig in Verbindung mit einem oder mehreren pharmazeutisch verträglichen Trägern oder Exzipienten. Ein pharmazeutisch verträglicher Träger oder Exzipient kann ein festes, halbfestes oder flüssiges Material sein, das als Vehikulum oder Medium für die Verbindung der Formel dient. Geeignete pharmazeutisch verträgliche Träger oder Exzipienten sind dem Fachmann gut bekannt.
- Eine pharmazeutische Zusammensetzung einer Verbindung der Formel kann entsprechend dem Verabreichungsweg angepasst werden. Eine bevorzugte pharmazeutische Zusammensetzung einer Verbindung der Formel ist eine Tablette, eine Pastille, eine Kapsel, ein Elixier, ein Saft, eine Oblate, ein Kaugummi, ein Zäpfchen, eine Lösung oder eine Suspension, wenn der Verabreichungsweg oral, parenteral oder oberflächlich ist.
- Eine bevorzugte orale pharmazeutische Zusammensetzung der Verbindung der Formel umfasst eine Verbindung der Formel mit einem inerten Verdünnungsmittel oder mit einem essbaren Träger. Bevorzugte Formen oraler pharmazeutischer Zusammensetzungen einer Verbindung der Formel sind Tabletten, Pastillen, Kapseln, Elixiere, Säfte, Oblate, Kaugummis, Zäpfchen, Lösungen oder Suspensionen.
- Bevorzugte pharmazeutische Zusammensetzungen der Formel enthalten etwa 4% bis etwa 80% der Verbindung. Bevorzugte pharmazeutische Zusammensetzungen enthalten eine Menge der Verbindung der Formel von etwa 50 ηg bis etwa 500 μg, bevorzugtere pharmazeutische Zusammensetzungen enthalten eine Menge der Verbindung der Formel von etwa 1 μg bis etwa 200 μg.
- Eine Verbindung der Formel kann einzeln oder in der Form einer pharmazeutischen Zusammensetzung in Kombination mit pharmazeutisch verträglichen Trägern oder Exzipienten verabreicht werden.
- Die nachfolgenden Abkürzungen werden hier verwendet:
mg, Milligramm; μg, Mikrogramm; ηg, Nanogramm; TEA, Triethylamin; mMol, Millimol; ml, Milliliter; °C, Celsius; h, Stunde; TLC. Dünnschichtchromatografie; CH2Cl2, Methylenchlorid; MeOH, Methanol; EtOH, Ethanol; N, Normal; HCl, Salzsäure; TFA, Trifluoressigsäure, DIEA, Diisopropylethylamin; RT-PCR, reverse Transkription-Polymerase-Kettenreaktion; HEPES, 4-(2-Hydroxyethyl)-1-piperazinethansulfonsäure; MgCl2; Magnesiumchlorid; EGTA, Ethylenglykol-bis(β-aminoethylether)-N,N,N'N'-tetraessigsäure; EDTA, Ethylendiamintetraessigsäure; DTT, Dithiothreitol; MOI, Multiplizität der Infektion; NaF, Natriumfluorid; BSA, Rinderserumalbumin; p. o., oral; i. v., intravenös; s. c., subkutan. - Beispiel 57
- Assay der Cyclin-abhängigen Kinase 4
- Die IC50-Werte für die cdk4-Hemmung wurden gemäß des nachfolgenden Verfahrens bestimmt:
- Substrat
- Glutathion S-Transferase-Retinablastom-Fusionprotein (GST-Rb) (Kaelin, W. G., Jr., et al., Cell 64: 521–532, 1991) wurde von Dr. William Kaelin erhalten. GST-Rb wurde durch Transformation von E. coli mit dem Plasmid pGEX-Rb (379–928) hergestellt. Die transformierten Bakterien wurden über Nacht bis zur Sättigung kultiviert, anschließend mit YT-Nährlösung verdünnt und 2 h bei 37°C inkubiert. Das Protein wurde durch 3-stündige Inkubation mit 0,1 mM Isopropylthioglycosid induziert. Nach der Sedimenation mittels Zentrifugation wurden die Zellen durch Ultraschall in STE-Puffer (0,1 mM NaCl, 10 mM Tris, pH 8,0, 1 mM EDTA), enthaltend 10% Sarkosyl, lysiert. Feststoffe wurden durch Zentrifugation entfernt und das Lysat wurde mit Glutathion-Sepharose bei 4°C inkubiert. Die Kügelchen wurden mit Kinasepuffer gewaschen und anschließend wurde die Quantifizierung der durch SDS-PAGE aufgetrennten mit Coomassie-Blau gefärbten Proteine unter Verwendung eines Proteinstandards bekannter Konzentration durchgeführt.
- Expression von cdk4/Cyclin B in Insektenzellen
- Humane Cyclin-abhängige Kinase 4 (cdk4) wurde mittels RT-PCR unter Verwendung von degenerierten Primern kloniert, basierend auf der veröffentlichten Aminosäuresequenz (Matsushime, H., et al., Cell 71: 323–334, 1992). Die cDNA für humanes Cyclin D1 wurde mittels RT-PCR unter Verwendung genomischer DNA von MCF-7-Zellen kloniert. Die Sequenz stimmte mit der veröffentlichten Sequenz überein (Xiong, Y., et al., Cell, 65: 691–699, 1991). Sowohl die cDNAs für cdk4 als auch für Cyclin D1 wurde in pFastBac (Life Technologies) kloniert und rekombinante Bacmid-DNA, enthaltend die cDNAs, wurde durch ortsspezifische Transposition mittels des Bac-to-Bac Baculovirus-Expressionssystems, bezogen von Life Technologies (Katalog # 10359-016), hergestellt. Bacmid-DNA wurde zur Transfektion von Sf9-Insektenzellen zur Herstellung rekombinanter Viren verwendet. Nach der Plaque-Reinigung des Virus wurden die viralen Präparationen verstärkt, bis Stämme mit hohen Titern erhalten wurden. Es wurde nachgewiesen, dass die optimale Co-Expression der rekombinanten Proteine bei einem MOI von 0,1 sowohl für cdk4 als auch für Cyclin D1 72 h nach der Infektion erreicht ist.
- Die Lysate wurden durch 5-minütige Lyse der mit cdk4 und Cyclin D1 co-infizierten Sf9-Zellen bei 4°C in 50 mM HEPES, pH 7,5, 10 mM MgCl2, 1 mM DTT, 0,1, mM Phenylmethylsulfonylfluorid, 5 μg/ml Aprotinin und 5 μg/ml Leupeptin mittels eines PARR-Druckgefäßes bei 500 psi Stickstoffdruck hergestellt. Das unlösliche Material wurde bei 4°C durch 20-minütige Zentrifugation bei 10 000 × g sedimentiert. Dem Überstand wurde 10% Glycerin zugesetzt und er wurde in Aliquoten bei –80°C gelagert.
- Kinase-Assay
- Millipore Multiscreen 96-Well Filterplatten (0,65 μm Durapore Filter) werden mit 200 μl Kinasepuffer (50 mM HEPES, pH 7,5, 10 mM MgCl2, 1 mM EGTA) befeuchtet. Pro Well werden 50 μl an Glutathion-Sepharose-Kügelchen gebundenes GST-Rb (0,5 μg) zugesetzt und die Lösung wird durch Anlegen von Vakuum entfernt. Das Assay enthält 50 mM HEPES, pH 7,5, 10 mM MgCl2, 1 mM EDTA, 1 mM DTT, 1 mM EGTA, 10 mM β-Glycerophosphat, 0,1 mM Natrium-orthovanadat, 0,1 mM NaF, 0,25% BSA, 10 μM ATP und 0,25 μCi [γ33P]-ATP. Um das Assay zu initiieren, setze 0,1 μg cdk4/Cyclin D1 (Insekten-Zelllysat) hinzu. Inkubiere 30 min bei 37°C. Beende die Reaktion durch Filtration mit einem Vacuum-Manifold von Millipore. Wasche viermal mit TNEN (20 mM Tris, pH 8,0, 100 mM NaCl, 1 mM EDTA, 0,5% Nonidet P-40). Nach dem Trocknen der Platten bei Raumtemperatur wurden die Platten in Adapterplatten (Packard) gesetzt und jedem Well wurde 40 μl Microscint-O® (Packard) zugesetzt. Vor der Zählung in einem TopCount Szintillationszähler wurden die Platten mit Top-Seal A Film verschlossen.
- Die Ergebnisse sind in Tabelle 1 dargestellt.
- Beispiel 58
- cdk2 Hemmstudien
- Die IC50-Werte für die cdk4-Hemmung wurden gemäß dem nachfolgenden Verfahren bestimmt:
- Assay der Cyclin-abhängigen Kinase 4
- Substrat
- GST-RB wie vorstehend für cdk4/Cyclin D1 beschrieben.
- Expression von cdk2/Cyclin E in Insektenzellen
- Rekombinante Baculoviren für humane(s) cdk2 und Cyclin E wurden von Dr. David Morgan bei UC, Berkley, erhalten (Desai, D., et al., Molec. Biol. Cell, 3: 571–582, 1992). Die optimale Co-Expression in Insektenzellen bei MOI's von 0,1 und 1,0 für cdk2 bzw. Cyclin E wurde 72 h nach der Infektion erreicht.
- Kinase-Assay
- Die Assay-Bedingungen für cdk2/Cyclin E entsprachen denen für cdk4/Cyclin D1, einschließlich des Substrats. Die Konzentration an rekombinantem cdk2/Cyclin E in dem Assay betrug 0,1 μg pro 100 μl Assay. Die Inkubation erfolgte 30 min bei 37°C.
- Die Ergebnisse sind in Tabelle 1 dargestellt.
- Beispiel 59
- cdk7/Cyclin H Assay-Protokoll
- Substrat
- Peptidsubstrat H2N-RRR(YSPTSPS)4-COOH basierend auf der CTD-Sequenz von RNA-Polymerase II.
- Expression von cdk7/Cyclin H in Insektenzellen
- Humane cdk7 wurde durch reverse Transkriptions-PCR kloniert. Die Sequenz stimmte mit der von Tassan, J. P., et al., J. Biol. Chem., 127: 467–478, 1994 und Darbon, J. M., et al., Oncogene, 9: 3127–3138, 1994 beschriebenen überein. Die cDNA für Cyclin H wurde ebenfalls mittels reverse Transkriptions- PCR kloniert und die Sequenz stimmte mit der von Fisher & Morgan, Cell, 78: 713–724, 1994 überein. Rekombinante Bacmid-DNA und virale Stämme wurden wie vorstehend für cdk4 und Cyclin D1 beschrieben hergestellt. Die optimale Co-Expression bei MOI's von 1 und 2 für cdk7 bzw. Cyclin H wurde 48 h nach der Infektion erreicht.
- Kinase-Assay
- Das Assay misst die Phosphorylierung eines Peptidsubstrats (basierend auf der C-terminalen Domaine von RNA-Polymerase II) durch die Cyclin-abhängige Kinase 7, die durch Cyclin H aktiviert wird. [γ33P]-Phosphat wird durch das Enzym aus [γ33P]-ATP auf das Peptidsubstrat übertragen. Das Assay wird in 96-Well-Platten mit V-förmigen Boden durchgeführt und nach der Beendigung wird die Reaktionslösung in 96-Well-Multiscreen-Phosphocellulose-Filter-Platten von Millipore übertragen. Das Peptid verbleibt nach dem Waschen mit Phosphorsäurelösung auf der Phosphocellulose-Membran.
- Verfahren
- Das Enzym-Assay wird in 96-Well-Platten mit V-förmigem Boden in einem Gesamtvolumen von 100 μl durchgeführt.
- Das Assay enthält 15 μM ATP, 0,5 μCi [γ33P]-ATP, 50 mM HEPES, pH 7,5, 10 mM MgCl2, 1 mM EDTA, 1 mM DTT, 10 mM β-Glycerophosphat, 0,1 mM Natrium-orthovanadat, 0,1 mM NaF, 10 μM Peptidsubstrat. Um das Assay zu initiieren, werden 0,125 ng cdk7 und Cyclin H (Insekten-Zelllysat) zugesetzt. Es wird 5 min bei 24°C inkubiert. Die Reaktion wird durch Zugabe von 40 μl kalter 300 mM Phosphorsäure zu jeder Probe beendet. Der Inhalt der V-förmigen Wells wird anschließend in eine 96-Well-Phosphocellulose-Filter-Platte von Millipore überführt. Nach 15-minütigem Stehen bei Raumtemperatur wurde Vakuum an die Filterplatte angelegt und die Wells wurden 4 × mit 100 μl kalter 75 mM Phosphorsäure gewaschen. Nach der Entfernung des un teren Filterzubehörs wurden die Filter vollständig getrocknet, in Multiscreen Mikroplatten Adapter gesetzt und jedem Well wurden 40 μl Microscint O zugesetzt. Die Platten wurden mit Top-Seal A Film verschlossen und 15 min mit einem TopCount Szintillationszähler von Packard gezählt.
- Die Ergebnisse sind in Tabelle 1 dargestellt.
- Beispiel 60
- cdk1/Cyclin [33P]SPA-Assay-Protokoll
- Substrat
- In dem Assay wird ein biotinyliertes Peptidsubstrat (Biotin-PKTPKKAKKL) verwendet, das von der in vitro p34cdc2-Phosphorylierungsstelle von Histon H1 abgeleitet ist.
- Expression von cdk1/Cyclin B1 in Insektenzellen
- Humane cdk1 wurde durch reverse Transkriptions-PCR kloniert. Die Sequenz stimmte mit der von Lee, M. G, und Nurse, P., Nature, 327: 31–33, 1987 überein. Die cDNA für Cyclin H wurde ebenfalls mittels reverser Transkriptions-PCR kloniert und die Sequenz stimmte mit der von Pines, J. und Hunter, T., Cell, 58: 833–846, 1989 überein. Rekombinante Bacmid-DNA und virale Stämme wurden wie vorstehend für cdk4 und Cyclin D1 beschrieben hergestellt. Die optimale Co-Expression bei einem MOI von 0,1 sowohl für cdk1 als auch für Cyclin B1 wurde 48 h nach der Infektion erreicht.
- Kinase-Assay
- Der p34cdc2 SPA[33P]-Kinase-Enzym-Assaykit wurde von Amersham Life Science (Katalog #RPNQ0170) bezogen und das Protokoll wurde, wie von den Herstellern vorgeschlagen, in einem 96-Well-Format durchgeführt. Jedes Assay enthielt 50 mM Tris, pH 8,0, 10 mM MgCl2, 0,1 mM Na3VO4 (Natrium-orthovanadat), 0,5 μM ATP, 0,2 μCi 33P-ATP, 2 μM DTT und 0,75 μM biotinyliertes Peptid und 3 μg cdk1/Cyclin B Insekten-Zelllysat in einem Ge samtvolumen vom 100 μl. Es wurde 30 min bei 30°C inkubiert. Die Reaktion wurde durch Zugabe von 200 μl Stop-Puffer (50 μM ATP, 5 mM EDTA, 0,1% (v/v) Triton X-100 in phosphatgepufferter Kochsalzlösung)/Streptavidin-beschichteten SPA-Kügelchen (2,5 mg/ml) beendet. Die Platte wurde über Nacht bei Raumtemperatur stehen lassen und anschließend mit einem TopSeal von Packard verschlossen und mittels eines TopCount von Packard gezählt. Die IC50-Werte wurden bestimmt, indem die Daten unter Nutzung von GraphPad Prism Software in eine sigmoidale Kurve angepasst wurden.
- Beispiel 61
- In vitro Tumor-Hemmung
- in vitro-Proliferationsassay
- Die Proliferation von Tumorzellen wurde mittels eines Sulforhodamin B-Assays bestimmt, wie in Shekan, P. et al., J. Natl. Cancer Inst., 82: 1107–1112, 1990 beschrieben. Die Tumorzellen wurden mit Trypsin-EDTA geerntet, die Zellen, die kein Tryphan-Blau aufnahmen, wurden gezählt, in 96-Well-Platten gegeben und über Nacht bei 37°C inkubiert. Der Arzneistoff wurde den Wells nach Verdünnen in Kulturmedium zugesetzt. Drei Tage später wurde das Medium entfernt und mit Medium, das frischen Arzneistoff enthielt, wieder aufgefüllt und weitere 4 Tage inkubiert. Anschließend wurden die Zellen mit 0,1 ml 10%iger Trichloressigsäure 60 min bei 4°C fixiert. Die Platten wurden 5-mal mit Leitungswasser gewaschen, an der Luft getrocknet und über einen Zeitraum von 30 min mit 0,4% Sulforhodamin B in 1% Essigsäure gefärbt und an der Luft getrocknet. Der gebundene Farbstoff wurde mit 0,1 ml 10 mM Tris (pH 10,5) 5 min solubilisiert und die Absorption wurde bei 490 nm mittels eines Titertek-Multiscan-MCC/340 Plattenreaders bestimmt.
- Alternativ wurde das CyQUANT-Zellproliferations-Assay zur Quantifizierung der Zellproliferation verwendet.
- CyQUANT-Zellproliferations-Assay
- Alternativ wurde das CyQUANT-Zellproliferations-Assay zur Quantifizierung der Tumorzellproliferation verwendet. Die Tumorzellen wurden mit Trypsin-EDTA geerntet, die Zellen, die kein Tryphan Blau aufnahmen wurden gezählt, in 96-Well-Platten gegeben und über Nacht bei 37°C inkubiert. Der Arzneistoff wurde den Wells nach Verdünnen in Kulturmedium zugesetzt. Drei Tage später wurde das Medium entfernt und die Platten wurden mindestens 30 Minuten bei –80°C eingefroren. Nach dem Auftauen der Platten wurde jedem Well 200 μl CyQUANT-GR in Zelllysepuffer (Molecular Probes # C-7026) zugesetzt und sie wurden 3–5 Minuten bei Raumtemperatur inkubiert. Die Fluoreszenz von CyQUANT-GR wurde mit einem Fmax-Fluoreszenz-Mikroplatten-Reader von Molecular Devices gemessen (Anregung 485 nm, Emission 530 nm).
- Zelllinien
-
- MCF7 ist eine humane Brust-Adenokarzinom-Zelllinie, Hormon-abhängig (HTB 22);
- MDA-MB-231 ist eine humane Brust-Adenokarzinom-Zelllinie, Hormon-unabhängig (HTB 26);
- HT-29 ist eine humane Kolon-Adenokarzinom-Zelllinie, mittlerer Differenzierungsgrad II (HTB 38);
- HCT-15 ist eine humane Kolon-Adenokarzinom-Zelllinie (CCL 225);
- A549 ist eine humane nicht-kleinzellige Lungen-Karziniom-Zelllinie (CCL 185);
- PC-3 ist eine humane Prostata-Adenokarzinom-Zelllinie, Hormon-unabhängig (CRL 1435); und
- DU 145 ist eine humane Prostata-Adenokarzinom-Zellinie, Hormon-unabhängig (HTB 81).
- Alle Zelllinien wurden von American Type Tissue Collection erhalten, die Hinterlegungsnummern sind in den Klammern angegeben.
- MCF-7-, MDA-MB-435- und MDA-MB-231-Zellen wurden in verbessertem Minimum-Essential-Medium (Biofluids) ohne Phenolrot, ergänzt mit 5% fötalem Kalbsserum, 0,01 mg/ml Gentamycin und 3 mM L-Glutamin, kultiviert. Alle anderen Zelllinien wurden in RPMI 1640 (Life Technologies), ergänzt mit 5% fötalem Kalbsserum, 0,01 mg/ml Gentamycin und 3 mM L-Glutamin, kultiviert.
- Die Ergebnisse sind in Tabelle 1 dargestellt.
- Beispiel 62
- in vivo-Assay
- Verfahren für das subrenale Kapsel-Assay
- Nacktmäuse wurden in Mikroisolator-Käfigen bei positivem Luftdruck gehalten. Die Operationsverfahren und Dosierungen wurden in einer Laminarbox durchgeführt. Das subrenale Kapsel-Assay wurde durchgeführt wie beschrieben von Bogden, A. E., Kelton, D. E., Cobb, W. R. und Esber, H. J.: A rapid screening method for testing chemotherapeutic agents against human tumor xenografts. In: D. P. Houchens und A. A. Ovejera (Hrsg.), Proceedings of the Symposium on the Use of Athymic (Nude) Mice in Cancer Research, S. 231–250. New York: Gustav Fischer New York, Inc. 1978. Kurz dargestellt wurden Tumore von 400–500 mm3, die von gehaltenen Mäusen erhalten wurden, in 1 mm3 Stücke geschnitten und die Größe wurde mittels eines Mikrometer-Okulars und eines Mikrotoms überprüft. Nach der Entnahme der Niere wurden die Stücke unter die Kapsel der Nieren der männlichen Nacktmäuse (6–8 Wochen alt) mittels eines 13 Ga Trochars implantiert. Nach der Implantation wurden die größten und kleinsten Durchmesser der Tumorstücke mittels eines Mikrometer-Okulars und eines Mikrotoms gemessen. Die Niere wurde in die Körperöffnung zurückgelegt und der Schnitt in der Körperwand wurde mittels chirugischem Nahtmaterial aus Seide verschlossen und der Hautschnitt wurde mit Clips verschlossen. Mit der Arzneistoffbehandlung wurde am Tag nach der Implantation begonnen und über einen Zeitraum von 12 Tagen fortgesetzt, wonach die Nieren erneut entnommen wurden und die Größe des Tumors wie vorstehend beschrieben bestimmt wurde. Das Tumorvolumen wurde gemäß der Formel V = Länge × Breite2/2 bestimmt, wie beschrieben in Houchens, D. P., Ovejera, A. A. und Barker, A. D. The therapy of human tumors in athymic (nude) mice. In: Proceedings of the Symposium on the Use of Athymic (Nude) Mice in Cancer Research, S. 267–280. New York: Gustav Fischer New York, Inc. 1978. Die Ergebnisse sind in Tabelle 2 dargestellt.
Claims (30)
- Verbindung der Formel worin R aus R2, R2NH- oder H2N-R3- ausgewählt ist, worin R2 aus C1-C8-Alkyl und ausgewählt ist, Z aus Phenyl, einem Heterocyclus, ausgewählt aus Piperidinyl, Pyridinyl, Isoxazolyl, Tetrahydrofuranyl, Pyrrolidinyl, Morpholinyl, Piperazinyl, Benzimidazolyl, Thiazolyl, Thiophenyl, Furanyl, Indolyl, 1,3-Benzodioxolyl, Tetrahydropyranyl, Imidazolyl, Tetrahydrothiophenyl, Pyranyl, Dioxanyl, Pyrrolyl, Pyrimidinyl, Pyrazinyl, Triazinyl, Oxazolyl, Purinyl, Chinolinyl und Isochinolinyl, und Cycloalkyl, ausgewählt ist jedes R4 unabhängig Wasserstoff oder C1-C4-Alkyl darstellt und n eine ganze Zahl von 1–8 ist, worin jedes C1-C8-Alkyl und Z gegebenenfalls mit 1 bis 3 Substituenten substituiert ist, die gleich oder verschieden sein können und aus Hal, OH, C1-C4-Alkyl ausgewählt sind; R3 C1-C8-Alkylen darstellt und R1 aus Cyclopentyl und Isopropyl ausgewählt ist und die pharmazeutisch verträglichen Salze, optischen Isomeren und Hydrate davon.
- Verbindung nach Anspruch 1, worin R R2 darstellt, wobei R2 C1-C8-Alkyl, gegebenenfalls substituiert mit 1 bis 3 Substituenten, die gleich oder verschieden sein können, ausgewählt aus Hal, OH und C1-C4-Alkyl, darstellt; R1 aus Cyclopentyl und Isopropyl ausgewählt ist und die pharmazeutisch verträglichen Salze, optischen Isomeren und Hydrate davon.
- Verbindung nach Anspruch 1, worin R R2 darstellt, wobei R2 darstellt, worin Z Phenyl, gegebenenfalls substituiert mit 1 bis 3 Substituenten, die gleich oder verschieden sein können, und aus Hal, OH und C1-C4-Alkyl ausgewählt sind, darstellt; jedes R4 unabhängig Wasserstoff oder C1-C4-Alkyl darstellt und n eine ganze Zahl von 1–8 ist; R1 aus Cyclopentyl und Isopropyl ausgewählt ist und die pharmazeutisch verträglichen Salze, optischen Isomeren und Hydrate davon.
- Verbindung nach Anspruch 1, worin R R2 darstellt, wobei R2 darstellt, worin Z einen Heterocyclus, wie in Anspruch 1 definiert, gegebenenfalls substituiert mit 1 bis 3 Substituenten, die gleich oder verschieden sein können und aus Hal, OH und C1-C4-Alkyl ausgewählt sind, darstellt; jedes R4 unabhängig Wasserstoff oder C1-C4-Alkyl darstellt und n eine ganze Zahl von 1 bis 8 ist; R1 aus Cyclopentyl und Isopropyl ausgewählt ist und die pharmazeutisch verträglichen Salze, optischen Isomeren und Hydrate davon.
- Verbindung nach Anspruch 1, worin R R2 darstellt, wobei R2 darstellt, worin Z Cycloalkyl, gegebenenfalls substituiert mit 1 bis 3 Substituenten, die gleich oder verschieden sein können und aus Hal, OH und C1-C4-Alkyl ausgewählt sind, darstellt; jedes R4 unabhängig Wasserstoff oder C1-C4-Alkyl darstellt und n eine ganze Zahl von 1 bis 8 ist; R1 aus Cyclopentyl und Isopropyl ausgewählt ist und die pharmazeutisch verträglichen Salze, optischen Isomeren und Hydrate davon.
- Verbindung nach Anspruch 1, worin R R2NH- darstellt, worin R2 C1-C8-Alkyl, das gegebenenfalls mit 1 bis 3 Substituenten substituiert ist, die gleich oder verschieden sein können und die aus Hal, OH und C1-C4-Alkyl ausgewählt sind, darstellt; R1 aus Cyclopentyl und Isopropyl ausgewählt ist und die pharmazeutisch verträglichen Salze, optischen Isomeren und Hydrate davon.
- Verbindung nach Anspruch 1, worin R R2NH- darstellt, wobei R2 darstellt, worin Z Phenyl, gegebenenfalls substituiert mit 1 bis 3 Substituenten, die gleich oder verschieden sein können und aus Hal, OH und C1-C4-Alkyl ausgewählt sind, darstellt; jedes R4 unabhängig Wasserstoff oder C1-C4-Alkyl darstellt und n eine ganze Zahl von 1 bis 8 ist; R1 aus Cyclopentyl und Isopropyl ausgewählt ist und die pharmazeutisch verträglichen Salze, optischen Isomeren und Hydrate davon.
- Verbindung nach Anspruch 1, worin R R2NH- darstellt, wobei R2 darstellt, worin Z einen Heterocyclus, wie in Anspruch 1 definiert, gegebenenfalls substituiert mit 1 bis 3 Substituenten, die gleich oder verschieden sind und aus Hal, OH und C1-C4-Alkyl ausgewählt sind, darstellt; jedes R4 unabhängig Wasserstoff oder C1-C4-Alkyl darstellt und n eine ganze Zahl von 1 bis 8 ist; R1 aus Cyclopentyl und Isopropyl ausgewählt ist und die pharmazeutisch verträglichen Salze, optischen Isomeren und Hydrate davon.
- Verbindung nach Anspruch 1, worin R R2NH- darstellt, wobei R2 darstellt, worin Z Cycloalkyl, gegebenenfalls substituiert mit 1 bis 3 Substituenten, die gleich oder verschieden sein können und aus Hal, OH und C1-C4-Alkyl ausgewählt sind, darstellt; jedes R4 unabhängig Wasserstoff oder C1-C4-Alkyl darstellt und n eine ganze Zahl von 1 bis 8 ist; R1 aus Cyclopentyl und Isopropyl ausgewählt ist und die pharmazeutisch verträglichen Salze, optischen Isomeren und Hydrate davon.
- Verbindung nach Anspruch 9, worin R H2N-R3- darstellt, worin R3 C1-C8-Alkylen darstellt und R1 aus Cyclopentyl und Isopropyl ausgewählt ist und die pharmazeutisch verträglichen Salze, optischen Isomeren und Hydrate davon.
- Verbindung nach einem der Ansprüche 1 bis 10, die ausgewählt ist aus 2-[trans-(4-Aminocyclohexyl)amino]-6-butylamino-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-octylamino-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-(3-methyl-4-hydroxy)butyl)amino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(5-hydroxy-1,5-dimethylhexyl)amino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[5-(hydroxy)pentylamino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-hydroxypropyl)amino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-(pentylamino)-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-(methylamino)-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-(ethylamino)-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-methylbutyl)-amino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(butyl)-amino]-9-(2-propyl)purindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-(hexylamino)-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-(propylamino)-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[2,2,2-trifluorethylamino]-9-cyclopentylpurindihydrochlorid, (S)-2-[trans-(4-Aminocyclohexyl)amino]-6-[(α-methylbenzyl)amino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-(3-jodbenzylamino)-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3,4-dichlorbenzyl)amino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-methylbenzyl)-amino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-chlor)-benzylamino]-9-(2-propyl)purindihydrochlorid, (R)-2-[trans-(4-Aminocyclohexyl)amino]-6-[(α-methylbenzyl)amino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(2-chlorbenzyl)-amino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-(4-phenylbutylamino)-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[3-(phenyl)-propylamino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-chlorbenzyl)amino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-chlorbenzyl)amino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-chlor-2-fluorbenzyl)amino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-hydroxyphenyl)ethylamino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-(benzylamino)-9-(2-propyl)purintrihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[benzylamino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(2,4-dichlorbenzyl)amino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-fluorbenzyl)-amino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-jodbenzyl)-amino]-9-(2-propyl)purindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-indolyl)-2-ethylamino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[2-(3,4-methylendioxyphenyl)ethylamino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-pyridyl)-2-ethylamino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-pyridyl)-methylamino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[3-(4-morpholinyl)propylamino]-9-cyclopentylpurintrihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[2-(2-pyridyl)ethylamino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[2-(4-morpholinyl)ethylamino]-9-cyclopentylpurintrihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-(2-thiophenmethylamino)-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(2-benzimidazolyl)methylamino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(2-tetrahydropyranyl)methylamino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-pyridyl)-2-ethylamino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-(2-furanmethylamino)-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(3-(2-imidazoyl)-propyl)amino]-9-(2-propyl)purintrihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-(3-(1-imidazolyl)-propylamino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[3-(2-imidazolyl)-propylamino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(cyclohexyl)-methylamino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(cyclopropyl)-methylamino]-9-(2-propyl)purindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(cyclopropyl)-methylamino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[2,2,2-trifluorethylhydrazino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[2-hydroxyethylhydrazino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[2-phenethylhydrazino]-9-cyclopentylpurindihydrochlorid, 2-[trans-(4-Aminocyclohexyl)amino]-6-[(4-aminobutyl)amino]-9-cyclopentylpurindihydrochlorid und 2-[trans-(4-Aminocyclohexyl)amino]-6-[(8-aminooctyl)amino]-9-cyclopentylpurindihydrochlorid.
- Verbindung nach einem der Ansprüche 1 bis 11 zur Verwendung als Arzneimittel.
- Pharmazeutische Zusammensetzung, enthaltend eine Verbindung nach einem der Ansprüche 1 bis 11 in Anmischung mit einem pharmazeutisch verträglichen Träger.
- Verwendung einer Verbindung nach einem der Ansprüche 1 bis 11 zur Herstellung eines Arzneimittels zur Behandlung einer hyperproliferativen Störung.
- Verwendung nach Anspruch 14, wobei die hyperproliferative Störung ein neoplastischer Krankheitszustand ist.
- Verwendung nach Anspruch 15, wobei der neoplastische Krankheitszustand aus Leukämie, Karzinom, Adenokarzinom, Sarkom, Melanom oder einem Neoplasmamischtyp ausgewählt ist.
- Verwendung nach Anspruch 16, wobei die Leukämie aus akuter lymphoblastischer Leukämie, chronischer Leukämie, akuter myeloblastischer Leukämie und chronischer myeloblastischer Leukämie ausgewählt ist.
- Verwendung nach Anspruch 16, wobei das Karzinom aus jenen von Gebärmutterhals, Brust, Prostata, Ösophagus, Magen, Dünndarm, Kolon, Ovarium und Lunge ausgewählt ist.
- Verwendung nach Anspruch 16, wobei das Adenokarzinom aus jenen von Gebärmutterhals, Brust, Prostata, Ösophagus, Magen, Dünndarm, Kolon, Ovarium und Lunge ausgewählt ist.
- Verwendung nach Anspruch 16, wobei das Sarkom aus Österom, Osteosarkom, Lipom, Liposarkom, Hämangiom und Hämangiosarkom ausgewählt ist.
- Verwendung nach Anspruch 16, wobei das Melanom aus amelanotischem Melanom und melanotischem Melanom ausgewählt ist.
- Verwendung nach Anspruch 16, wobei der Neoplasmamischtyp aus Carcinosarkom, lymphoidem Gewebstyp, folikullärem Retikulum, Zellsarkom und Hodgkinscher Krankheit ausgewählt ist.
- Verwendung einer Verbindung nach einem der Ansprüche 1 bis 11 zur Herstellung eines Arzneimittels zum Verhindern von Apoptosis in neuronalen Zellen.
- Verwendung einer Verbindung nach Anspruch 23, wobei die Apoptosis durch ein antineoplastisches Mittel induziert wird.
- Verwendung einer Verbindung nach Anspruch 23, wobei die Apoptosis durch eine cerebrovaskuläre Erkrankung induziert wird.
- Verwendung einer Verbindung nach Anspruch 23, wobei die Apoptosis durch Schlaganfall oder Infarkt induziert wird.
- Verwendung einer Verbindung nach einem der Ansprüche 1 bis 11 zum Herstellen eines Arzneimittels zum Schutz von neuronalen Zellen gegen Apoptosis.
- Verwendung einer Verbindung nach einem der Ansprüche 1 bis 11 zur Herstellung eines Arzneimittels zum Schutz von neuronalen Zellen gegen Schädigung, die durch ein antineoplastisches Mittel induziert wird.
- Zusammensetzung, umfassend eine bestimmbare Menge einer Verbindung nach einem der Ansprüche 1 bis 11 in Anmischung oder anderweitig in Verbindung mit einem inerten Träger.
- Pharmazeutische Zusammensetzung, umfassend eine wirksame cdk-2 inhibierende Menge einer Verbindung nach einem der Ansprüche 1 bis 11 in Anmischung oder anderweitig in Verbindung mit einem oder mehreren pharmazeutisch verträglichen Trägern oder Exzipienten.
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US3238198A | 1998-02-26 | 1998-02-26 | |
| US32381 | 1998-02-26 | ||
| PCT/US1999/003451 WO1999043676A2 (en) | 1998-02-26 | 1999-02-18 | 6,9-disubstituted 2-[trans-(4- aminocyclohexyl) amino]purines |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| DE69918062D1 DE69918062D1 (de) | 2004-07-22 |
| DE69918062T2 true DE69918062T2 (de) | 2005-06-02 |
Family
ID=21864658
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| DE69918062T Expired - Fee Related DE69918062T2 (de) | 1998-02-26 | 1999-02-18 | 6,9-disubstituierte 2-(trans-(4-aminocyclohexyl)amino)purine |
Country Status (30)
| Country | Link |
|---|---|
| EP (1) | EP1056745B1 (de) |
| JP (1) | JP2002504553A (de) |
| KR (1) | KR100568658B1 (de) |
| CN (1) | CN1128149C (de) |
| AP (1) | AP1258A (de) |
| AR (1) | AR019822A1 (de) |
| AT (1) | ATE269332T1 (de) |
| AU (1) | AU748178B2 (de) |
| BR (1) | BR9908325A (de) |
| CA (1) | CA2320448C (de) |
| CZ (1) | CZ291604B6 (de) |
| DE (1) | DE69918062T2 (de) |
| DK (1) | DK1056745T3 (de) |
| EE (1) | EE04315B1 (de) |
| ES (1) | ES2219038T3 (de) |
| HU (1) | HUP0100931A3 (de) |
| IL (2) | IL138044A0 (de) |
| NO (1) | NO20004281L (de) |
| NZ (1) | NZ506234A (de) |
| OA (1) | OA11480A (de) |
| PL (1) | PL344021A1 (de) |
| PT (1) | PT1056745E (de) |
| RU (1) | RU2191777C2 (de) |
| SI (1) | SI1056745T1 (de) |
| SK (1) | SK12912000A3 (de) |
| TR (1) | TR200002482T2 (de) |
| TW (1) | TW541308B (de) |
| UA (1) | UA62996C2 (de) |
| WO (1) | WO1999043676A2 (de) |
| ZA (1) | ZA991548B (de) |
Families Citing this family (28)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US6969720B2 (en) | 1999-03-17 | 2005-11-29 | Amr Technology, Inc. | Biaryl substituted purine derivatives as potent antiproliferative agents |
| US6627633B2 (en) | 1999-03-17 | 2003-09-30 | Albany Molecular Research, Inc. | 6-substituted biaryl purine derivatives as potent cyclin/CDK inhibitors and antiproliferative agents |
| CA2396276A1 (en) * | 2000-01-07 | 2001-07-12 | Universitaire Instelling Antwerpen | Purine derivatives, process for their preparation and use thereof |
| YU54202A (sh) | 2000-01-18 | 2006-01-16 | Agouron Pharmaceuticals Inc. | Jedinjenja indazola, farmaceutske smeše i postupci za stimulisanje i inhibiranje ćelijske proliferacije |
| HN2001000008A (es) | 2000-01-21 | 2003-12-11 | Inc Agouron Pharmaceuticals | Compuesto de amida y composiciones farmaceuticas para inhibir proteinquinasas, y su modo de empleo |
| FR2806626B1 (fr) * | 2000-03-22 | 2003-11-28 | Centre Nat Rech Scient | Utilisation de substances modulatrices de l'expression ou de la fonction d'une proteine impliquee dans le cycle cellulaire pour le traitement ou la prevention des lesions neurales aigues |
| BR0110302A (pt) | 2000-04-18 | 2003-01-14 | Agouron Pharma | Compostos de pirazol para inibição de proteìnas cinase, sal e pró-droga farmaceuticamente aceitável, metabólito farmaceuticamente ativo ou sal farmaceuticamente aceitável de metabólito, composição farmacêutica, método de tratamento de condição doentia em mamìferos mediada pela atividade de proteìna cinase, método de modulação ou inibição da atividade de um receptor de proteìna cinase |
| EP1307462A2 (de) | 2000-08-09 | 2003-05-07 | Agouron Pharmaceuticals, Inc. | Pyrazol-thiazol-verbindungen, diese enthaltende pharmazeutische zusammensetzungen und methoden ihrer verwendung zur hemmung cyclin-abhängiger kinasen |
| JP2004506720A (ja) | 2000-08-18 | 2004-03-04 | アグーロン ファーマシューティカルズ,インコーポレイテッド | 複素環式ヒドロキシイミノフルオレン、およびプロテインキナーゼ阻害のためのこれらの使用 |
| GB0117075D0 (en) * | 2000-10-31 | 2001-09-05 | Aventis Pharm Prod Inc | Acyl and sulfonyl derivatives of 6, 9-distributed 2-(trans-1, 4-diaminocyclohexyl)-purines and their use as antiproliferative agents |
| DE60138140D1 (de) * | 2000-10-31 | 2009-05-07 | Aventis Pharma Inc | Acyl- und sulfonylderivative 6,9-disubstitutierter 2-(trans-1,4-diaminocyclohexyl)-purine und ihre verwendung als antiproliferative mittel |
| US6756374B2 (en) | 2001-01-22 | 2004-06-29 | Hoffmann-La Roche Inc. | Diaminothiazoles having antiproliferative activity |
| DE60205376T2 (de) | 2001-06-27 | 2006-04-06 | Cyclacel Ltd. | 2,6,9-substituierte purinderivate und ihre verwendung bei der behandlung proliferativer krankheiten |
| US6812232B2 (en) | 2001-09-11 | 2004-11-02 | Amr Technology, Inc. | Heterocycle substituted purine derivatives as potent antiproliferative agents |
| US6667311B2 (en) | 2001-09-11 | 2003-12-23 | Albany Molecular Research, Inc. | Nitrogen substituted biaryl purine derivatives as potent antiproliferative agents |
| GB0219054D0 (en) | 2002-08-15 | 2002-09-25 | Cyclacel Ltd | New purine derivatives |
| GB0219052D0 (en) | 2002-08-15 | 2002-09-25 | Cyclacel Ltd | New puring derivatives |
| JPWO2004069233A1 (ja) * | 2003-02-04 | 2006-05-25 | 株式会社ヤクルト本社 | 乳癌耐性蛋白阻害剤 |
| US7211576B2 (en) | 2004-04-20 | 2007-05-01 | Hoffmann-La Roche Inc. | Diaminothiazoles |
| SG170819A1 (en) * | 2006-03-30 | 2011-05-30 | Ptc Therapeutics Inc | Methods for the production of functional protein from dna having a nonsense mutation and the treatment of disorders associated therewith |
| MY150993A (en) * | 2007-10-05 | 2014-03-31 | Verastem Inc | Pyrimidine substituted purine derivatives |
| CN102241646B (zh) * | 2010-05-14 | 2014-04-09 | 中国人民解放军军事医学科学院毒物药物研究所 | 己烯酮类化合物及其医药用途 |
| US9493464B2 (en) | 2012-02-29 | 2016-11-15 | The Scripps Research Institute | Wee1 degradation inhibitors |
| JPWO2015005491A1 (ja) * | 2013-07-12 | 2017-03-02 | 国立大学法人京都大学 | 疾病の発症又は進行の一因となる異常スプライシングを抑制できる物質のスクリーニング方法 |
| EP3244891B9 (de) | 2015-01-16 | 2022-11-16 | The General Hospital Corporation | Verbindungen zur verbesserung des spleissens von mrna |
| CN107903274A (zh) * | 2017-12-28 | 2018-04-13 | 窦玉玲 | 一种胺类化合物及其在抗肿瘤药物中的应用 |
| WO2021016263A1 (en) * | 2019-07-21 | 2021-01-28 | University Of Virginia Patent Foundation | Cysteine binding compositions and methods of use thereof |
| WO2024105159A1 (en) * | 2022-11-16 | 2024-05-23 | University Of Zurich | Ligands of the m6a-rna readers |
Family Cites Families (4)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| GB9301000D0 (en) * | 1993-01-20 | 1993-03-10 | Glaxo Group Ltd | Chemical compounds |
| GB9414208D0 (en) * | 1994-07-14 | 1994-08-31 | Glaxo Group Ltd | Compounds |
| EP0874846B1 (de) * | 1995-11-01 | 2003-04-02 | Novartis AG | Purinderivate und verfahren zu ihrer herstellung |
| FR2741881B1 (fr) * | 1995-12-01 | 1999-07-30 | Centre Nat Rech Scient | Nouveaux derives de purine possedant notamment des prorietes anti-proliferatives et leurs applications biologiques |
-
1999
- 1999-02-18 RU RU2000124409/04A patent/RU2191777C2/ru active
- 1999-02-18 TR TR2000/02482T patent/TR200002482T2/xx unknown
- 1999-02-18 NZ NZ506234A patent/NZ506234A/en active IP Right Revival
- 1999-02-18 CN CN99803395A patent/CN1128149C/zh not_active Expired - Fee Related
- 1999-02-18 ES ES99936074T patent/ES2219038T3/es not_active Expired - Lifetime
- 1999-02-18 IL IL13804499A patent/IL138044A0/xx active IP Right Grant
- 1999-02-18 UA UA2000095500A patent/UA62996C2/uk unknown
- 1999-02-18 SK SK1291-2000A patent/SK12912000A3/sk unknown
- 1999-02-18 AT AT99936074T patent/ATE269332T1/de not_active IP Right Cessation
- 1999-02-18 PT PT99936074T patent/PT1056745E/pt unknown
- 1999-02-18 PL PL99344021A patent/PL344021A1/xx unknown
- 1999-02-18 DE DE69918062T patent/DE69918062T2/de not_active Expired - Fee Related
- 1999-02-18 EP EP99936074A patent/EP1056745B1/de not_active Expired - Lifetime
- 1999-02-18 SI SI9930621T patent/SI1056745T1/xx unknown
- 1999-02-18 WO PCT/US1999/003451 patent/WO1999043676A2/en not_active Ceased
- 1999-02-18 EE EEP200000501A patent/EE04315B1/xx not_active IP Right Cessation
- 1999-02-18 BR BR9908325-6A patent/BR9908325A/pt not_active IP Right Cessation
- 1999-02-18 AU AU32990/99A patent/AU748178B2/en not_active Ceased
- 1999-02-18 DK DK99936074T patent/DK1056745T3/da active
- 1999-02-18 HU HU0100931A patent/HUP0100931A3/hu unknown
- 1999-02-18 CZ CZ20003106A patent/CZ291604B6/cs not_active IP Right Cessation
- 1999-02-18 KR KR1020007009421A patent/KR100568658B1/ko not_active Expired - Fee Related
- 1999-02-18 CA CA002320448A patent/CA2320448C/en not_active Expired - Fee Related
- 1999-02-18 AP APAP/P/2000/001923A patent/AP1258A/en active
- 1999-02-18 JP JP2000533431A patent/JP2002504553A/ja active Pending
- 1999-02-24 TW TW088102781A patent/TW541308B/zh not_active IP Right Cessation
- 1999-02-25 ZA ZA9901548A patent/ZA991548B/xx unknown
- 1999-02-25 AR ARP990100782A patent/AR019822A1/es active IP Right Grant
-
2000
- 2000-08-23 IL IL138044A patent/IL138044A/en not_active IP Right Cessation
- 2000-08-25 NO NO20004281A patent/NO20004281L/no not_active Application Discontinuation
- 2000-08-25 OA OA1200000234A patent/OA11480A/en unknown
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| DE69918062T2 (de) | 6,9-disubstituierte 2-(trans-(4-aminocyclohexyl)amino)purine | |
| US6413974B1 (en) | 6,9,-disubstituted 2-[trans-(4-aminocyclohexyl) amino] purines | |
| EP1056744B1 (de) | 6,9-disubstituierte 2-[trans-(4-aminocyclohexyl)amino] purine | |
| DE69832715T2 (de) | Cyclin-abhängige-kinase inhibierende purinderivate | |
| JP4348081B2 (ja) | 6,9−二置換2−(トランス−1,4−ジアミノシクロヘキシル)−プリンのアシル及びスルホニル誘導体及び抗増殖剤としてのその使用 | |
| KR20070033962A (ko) | 증식성 질병의 치료에서 9h-퓨린-2,6-디아민 유도체의용도 및 신규 9h-퓨린-2,6-디아민 유도체 | |
| HUE032987T2 (en) | Dihydropyrazolopyrimidinone derivative | |
| JP2010516774A (ja) | マラリア原虫関連疾患を処置するためのキナーゼ阻害剤としてのプリン化合物および組成物 | |
| JP2005527565A (ja) | 新しいピラゾロ[4,3−d]ピリミジン、その調整方法、及び治療方法 | |
| JP2006511458A (ja) | アザプリン誘導体 | |
| US7429595B2 (en) | Acyl and sulfonyl derivatives of 6,9-disubstituted 2-(trans-1,4-diaminocyclohexyl)-purines and their use as antiproliferative agents | |
| EP1619196A1 (de) | Substituierte Pyrido[3',2':4,5]thieno[3,2-d]pyrimidine und Pyrido[3',2':4,5]furo[3,2-d]-pyrimidine zur Verwendung als Inhibitoren der PDA-4 und/oder TNF-alpha Freisetzung | |
| DE69912248T2 (de) | 6,9-disubstituierte 2-[trans-(4-aminocyclohexyl)amino] purine | |
| US6642231B2 (en) | 6,9-disubstituted 2-[trans-(4-aminocyclohexyl)amino] purines | |
| KR100853106B1 (ko) | 6,9-이치환된 2-(트랜스-1,4-디아미노사이클로헥실)-퓨린의 아실 및 설포닐 유도체 및 항증식제로서의 이들의 용도 | |
| MXPA00008376A (en) | 6,9-disubstituted 2-[trans-(4- aminocyclohexyl) amino]purines | |
| HK1030948B (en) | 6,9-disubstituted 2-[trans-(4-aminocyclohexyl) amino] purines |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| 8364 | No opposition during term of opposition | ||
| 8328 | Change in the person/name/address of the agent |
Representative=s name: PFENNING MEINIG & PARTNER GBR, 80339 MUENCHEN |
|
| 8328 | Change in the person/name/address of the agent |
Representative=s name: MAIWALD PATENTANWALTSGESELLSCHAFT MBH, 80335 MUENC |
|
| 8327 | Change in the person/name/address of the patent owner |
Owner name: AVENTIS HOLDINGS INC., GREENVILLE, DEL., US |
|
| 8339 | Ceased/non-payment of the annual fee |