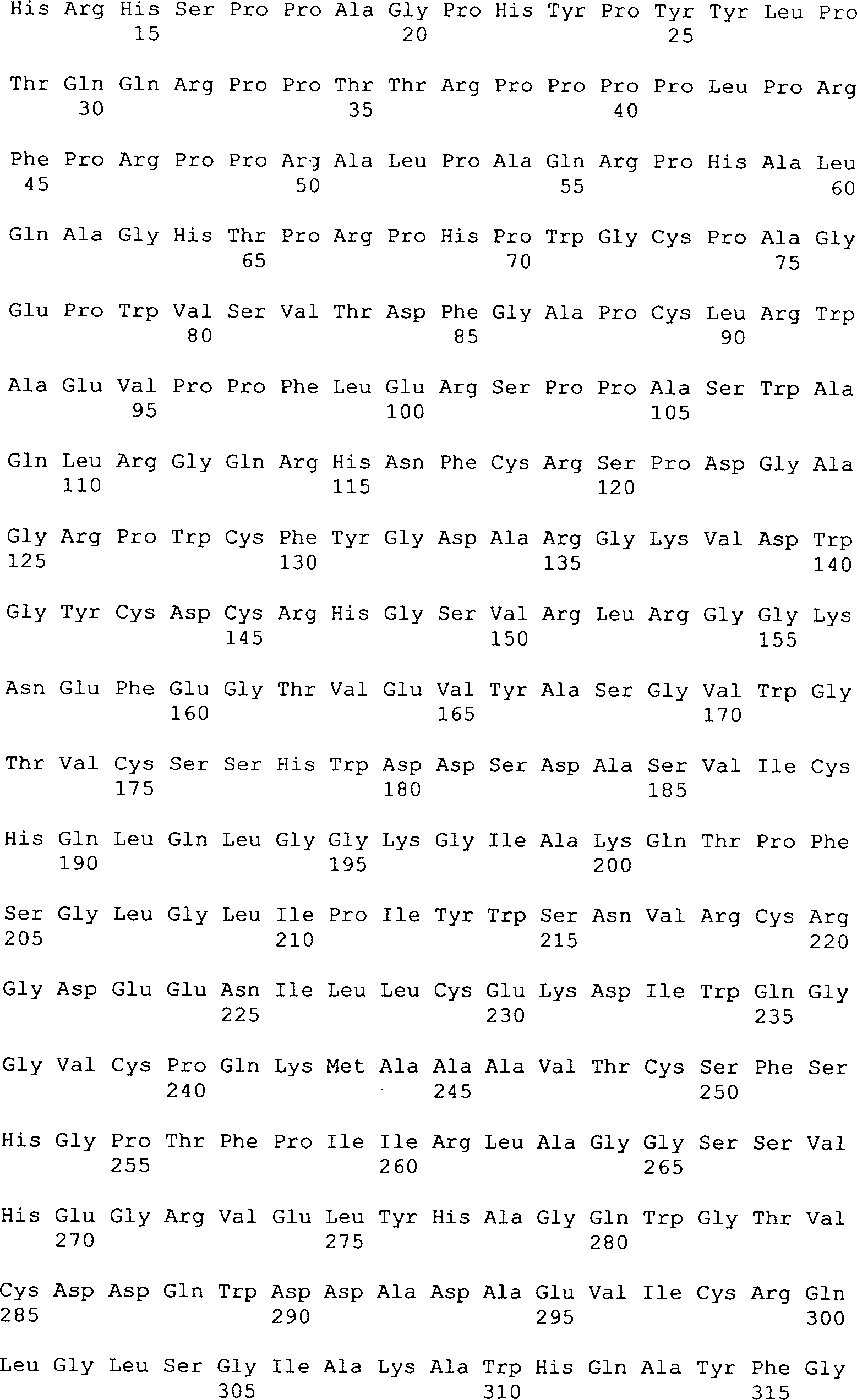-
Technisches
Gebiet
-
Die
vorliegende Erfindung betrifft Neurotrypsine und eine pharmazeutische
Zusammensetzung, welche diese Substanzen enthält oder auf diese Substanzen
einwirkt.
-
Beschreibung
der Erfindung
-
Neurotrypsin
ist eine neu entdeckte Serinprotease, welche vor allem im Gehirn
und in der Lunge exprimiert wird; die Expression im Gehirn findet
fast ausschliesslich in Nervenzellen statt.
-
Neurotrypsin
hat. eine bisher nicht gefundene Domänenzusammensetzung: neben der
Protease-Domäne
findet man 3 oder 4 SRCR (Scavenger Receptor Cysteine-Rich)-Domänen und
eine Kringel-Domäne. Es
ist hervorzuheben, dass die Kombination von Kringel- und SRCR-Domänen bisher
noch nie in Proteinen gefunden worden ist. Am Aminoterminus des
Neurotrypsin-Proteins befindet sich ein Segment von über 60 Aminosäuren, welches
einen ausserordentlich hohen Anteil von Prolin und basischen Aminosäuren (Arginin und
Histidin) aufweist.
-
Die
Erfindung ist durch die Merkmale in den unabhängigen Ansprüchen gekennzeichnet.
Bevorzugte Ausführungsformen
sind in den abhängigen
Ansprüchen
definiert.
-
Die
neu gefundenen Neurotrypsine
- – Neurotrypsin
des Menschen (Verbindung der Formel I),
- – Neurotrypsin
der Maus (Verbindung der Formel II)
unterscheiden sich
strukturell sehr stark von den bisher bekannten Serinproteasen.
-
Die
Serinprotease, deren Protease-Domäne strukturell am nächsten mit
der Protease-Domäne der neuen
Verbindungen verwandt ist, nämlich
Plasmin (des Menschen), weist eine Aminosäuresequenz-Identität von nur
44% auf.
-
Das
Prolin-reiche, basische Segment am Aminoterminus weist eine gewisse Ähnlichkeit
auf mit den basischen Segmenten der Netrine und der Semaphorine/Collapsine.
Aufgrund dieses Segmentes ist es wahrscheinlich, dass Neurotrypsin
mittels Heparin-Affinitätschromatographie
angereichert werden kann.
-
Die
Neurotrypsine des Menschen (Verbindung der Formel I) und der Maus
(Verbindung der Formel II) weisen unter sich eine sehr hohe strukturelle Ähnlichkeit
auf.
-
Die
Identität
der Aminosäuresequenzen
der nativen Proteine der Verbindungen der Formeln I oder II beträgt 81%.
-
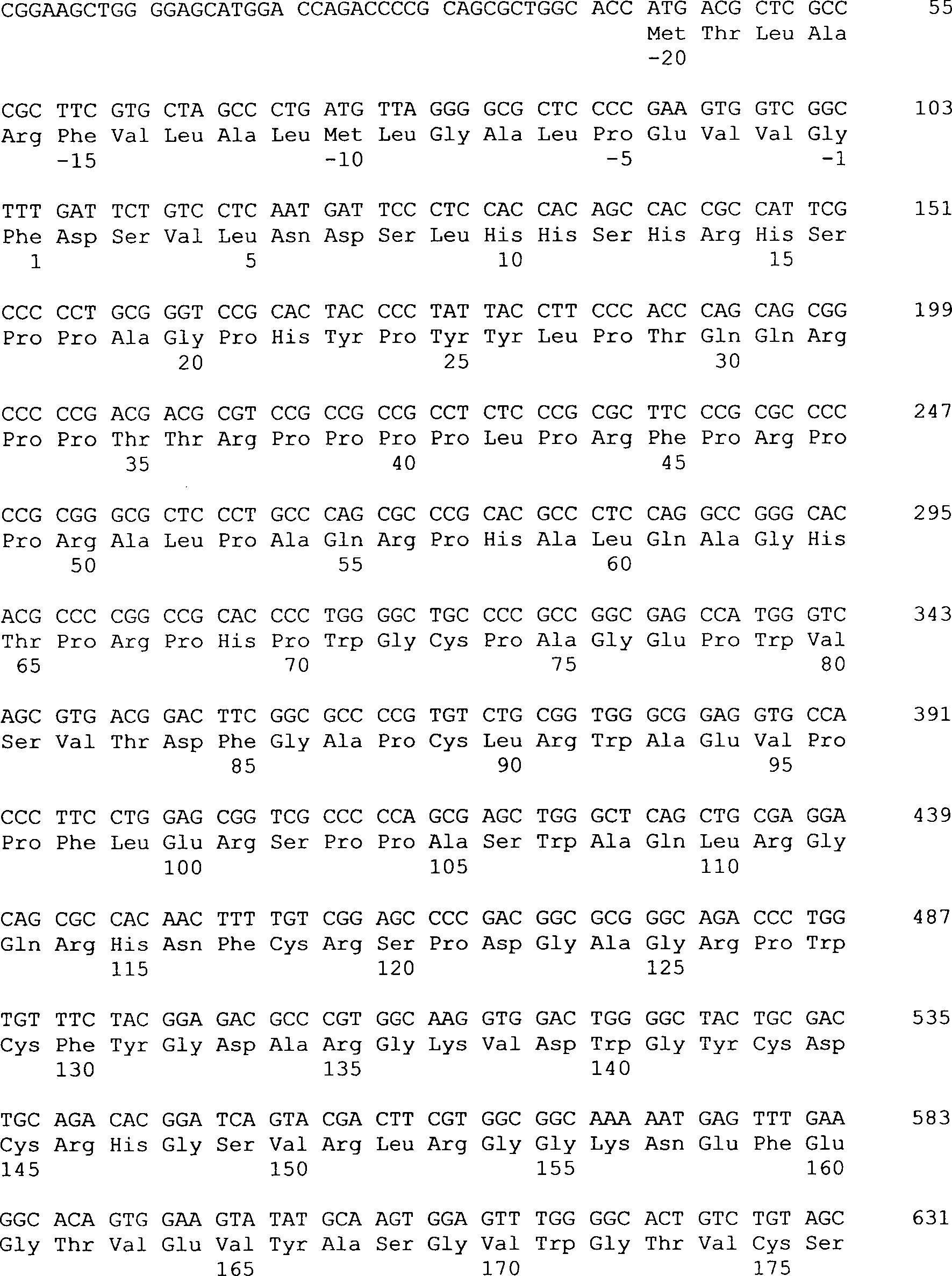
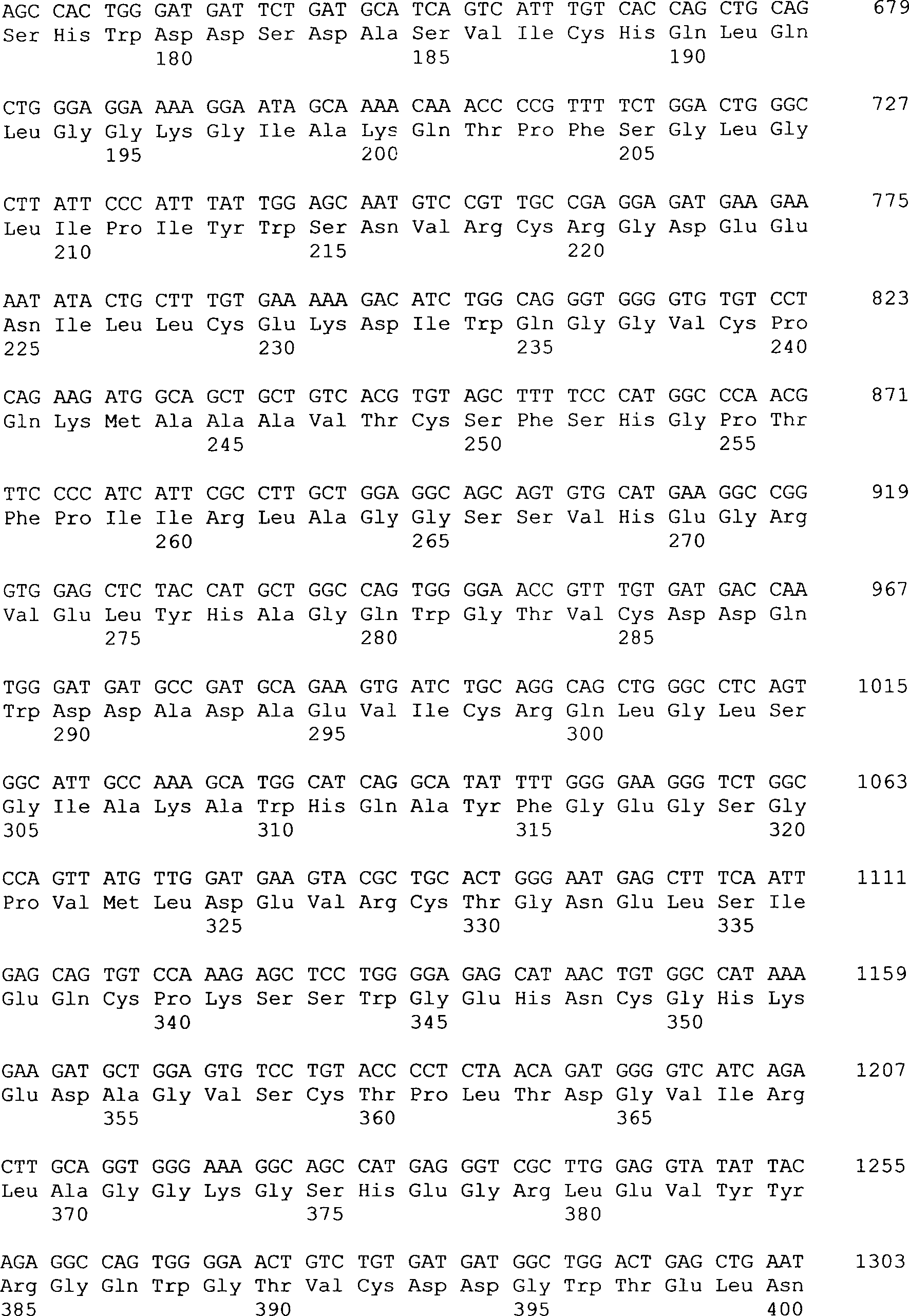
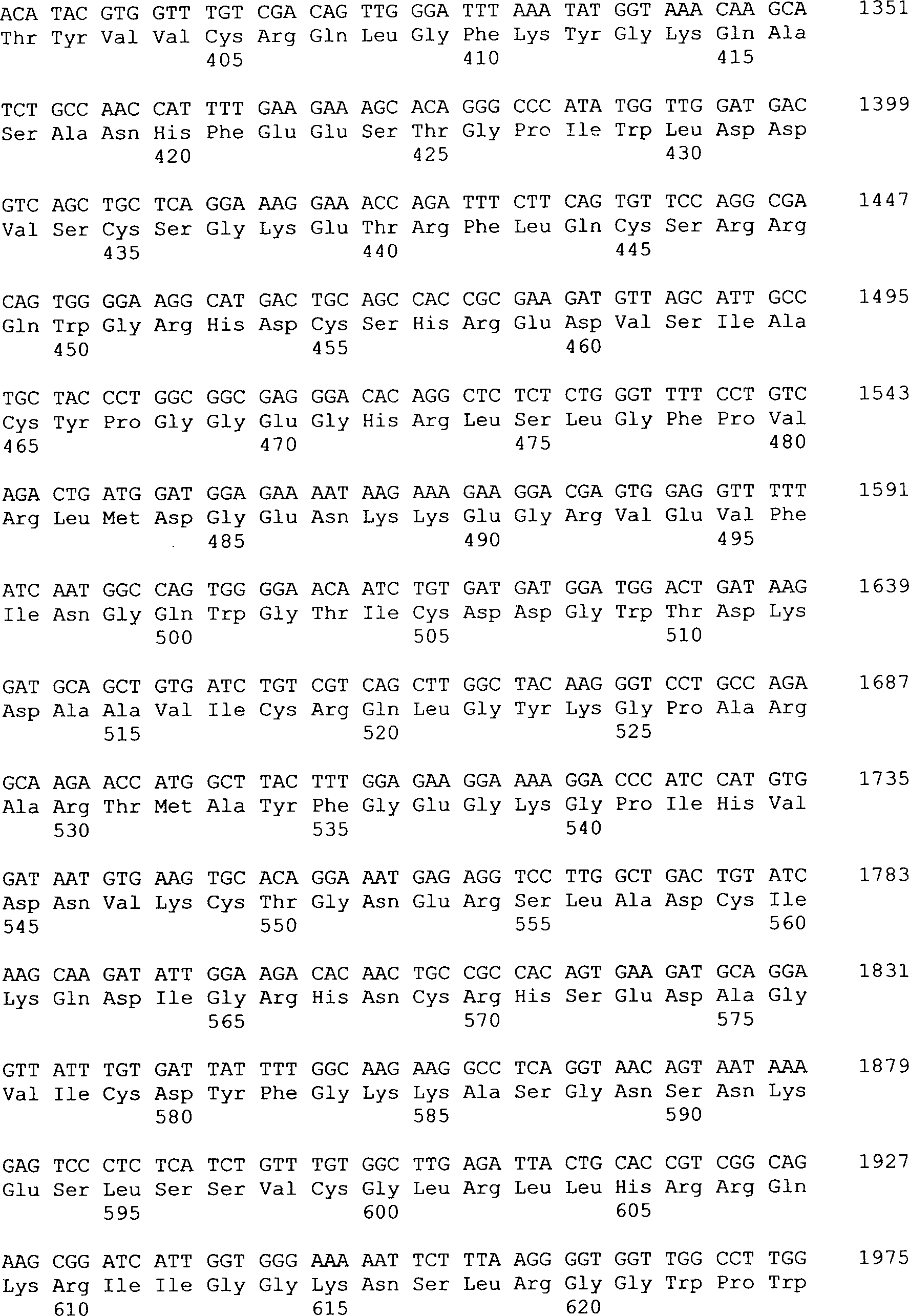
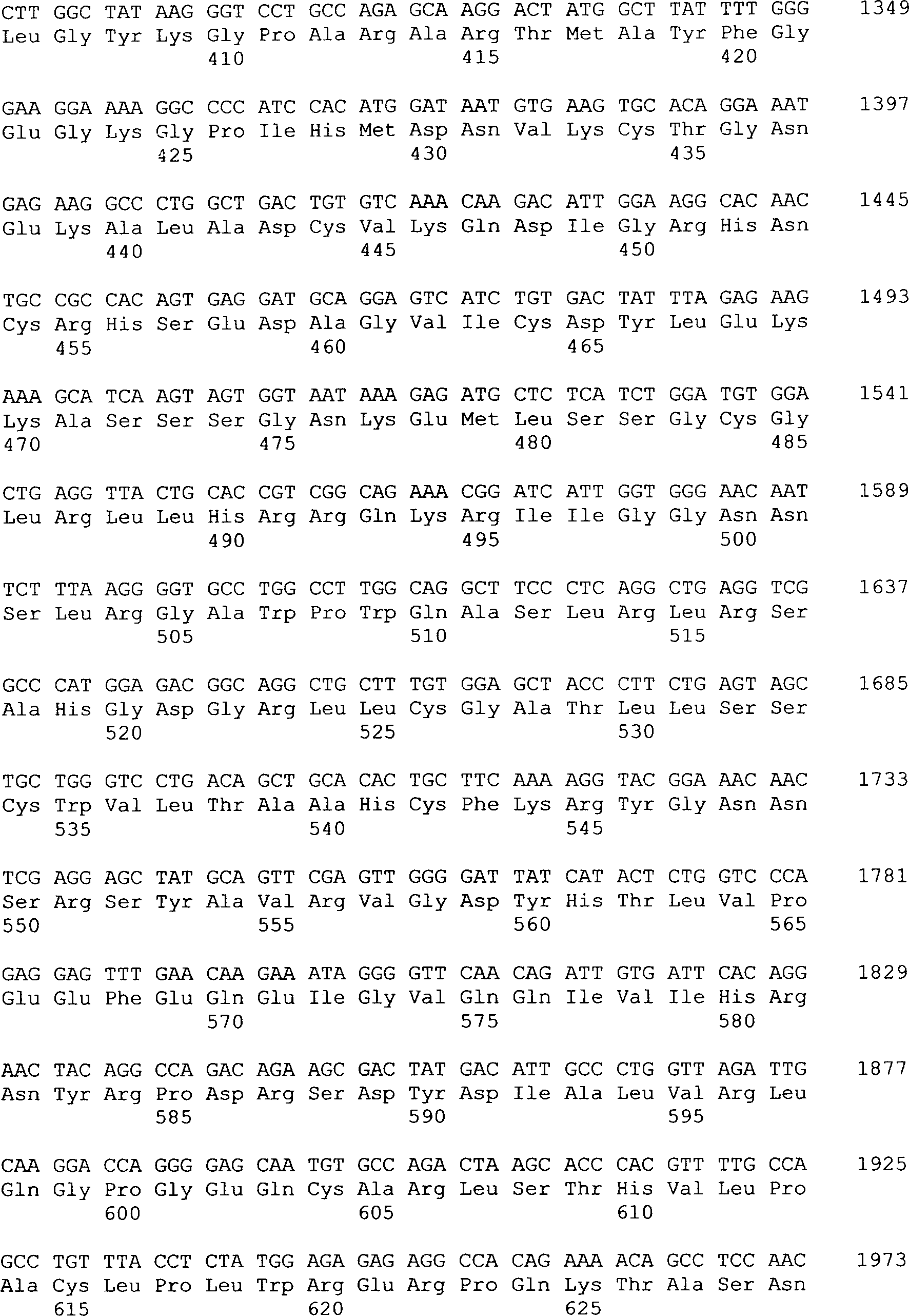
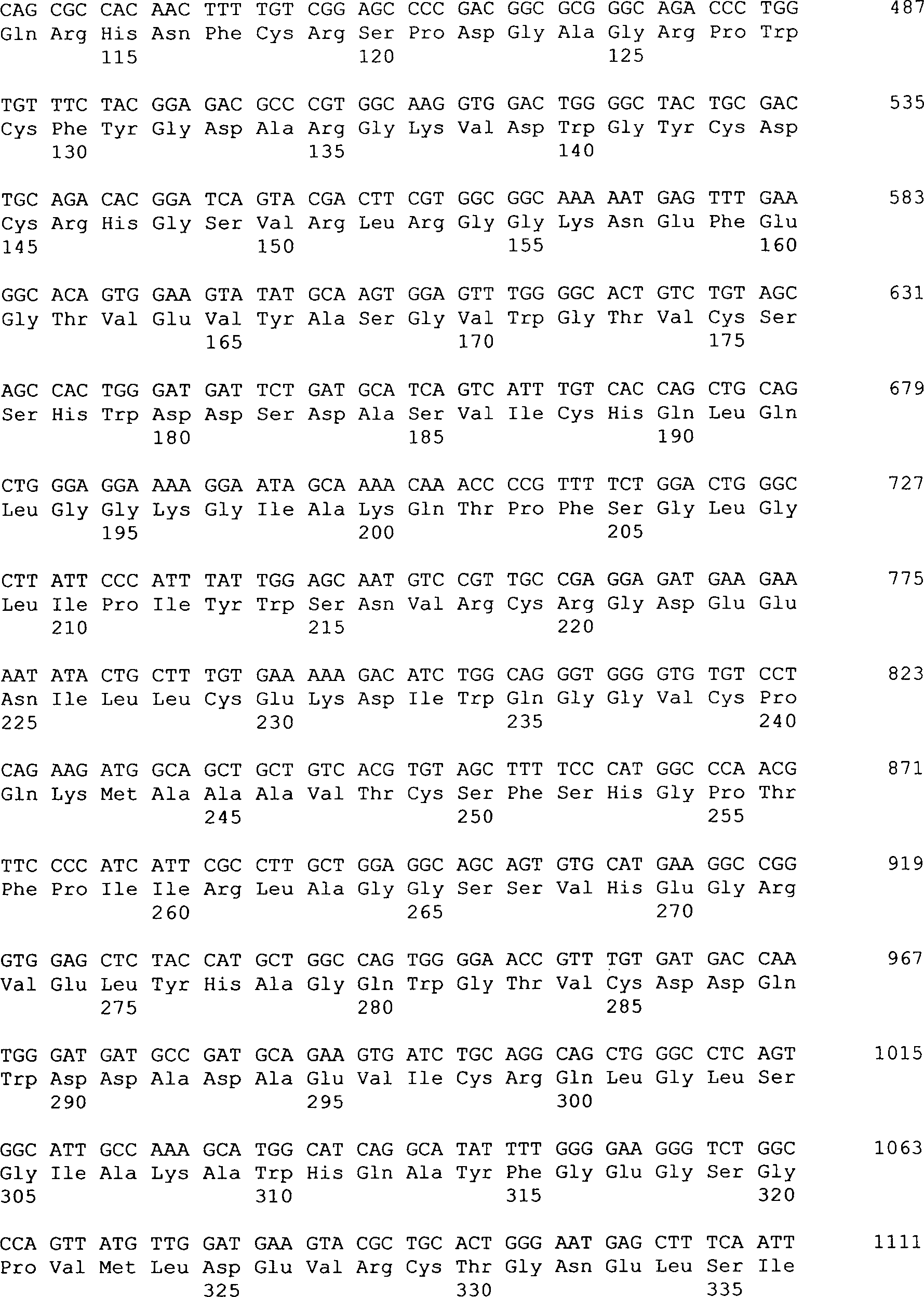
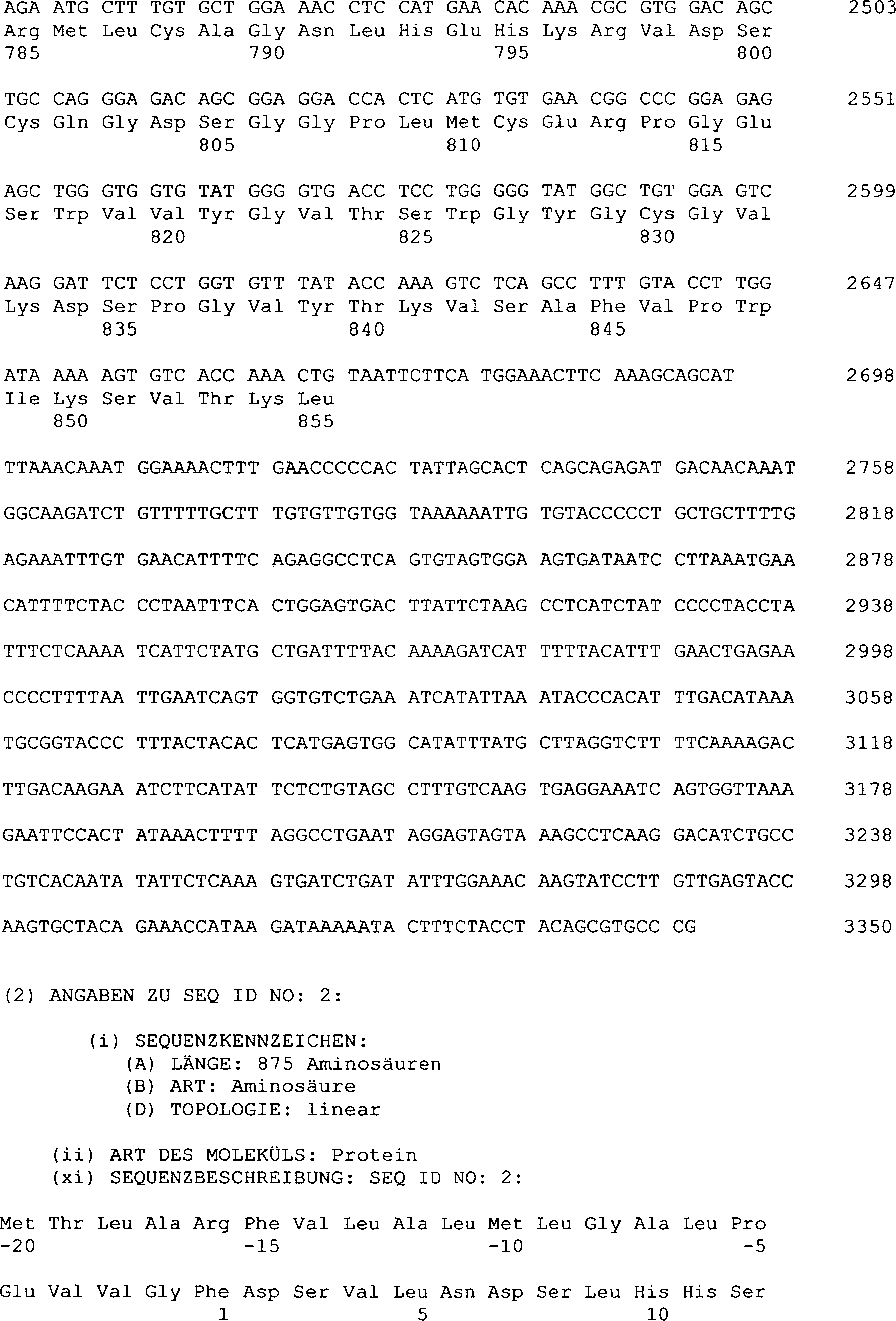
Das
Neurotrypsin des Menschen (Verbindung der Formel I) hat eine kodierende
Sequenz von 2625 Nukleotiden. Das kodierte Peptid der Verbindung
der Formel I ist 875 Aminosäuren
lang und enthält
ein Signalpeptid von 20 Aminosäuren.
Das Neurotrypsin der Maus (Verbindung der Formel II) hat eine kodierende Sequenz
von 2283 Nukleotiden. Das kodierte Protein der Verbindung der Formel
II ist 761 Aminosäuren
lang und enthält
ein Signalpeptid von 21 Aminosäuren.
Der Grund für
die grössere
Länge des
Neurotrypsins des Menschen liegt darin, dass das menschliche Neurotrypsin
4 SRCR-Domänen
aufweist, während
dem das Neurotrypsin der Maus nur 3 SRCR-Domänen hat.
-
Die
Domänen,
welche bei beiden Verbindungen (Verbindung der Formel I und Verbindung
der Formel II) vorhanden sind, weisen einen hohen Grad von Sequenzähnlichkeit
auf. Die einander entsprechenden SRCR-Domänen der Verbindungen der Formeln
I und II weisen eine Aminosäuresequenzidentität von 81%
bis 91% auf. Die entsprechenden Kringel-Domänen haben eine Aminosäuresequenzidentität von 75%.
Ein hoher Grad von Ähnlichkeit
besteht auch in der enzymatisch aktiven (d.h. proteolytischen) Domäne (90%
Aminosäuresequenzidentität).
-
Die
Proteasedomänen
der Neurotrypsine des Menschen (Verbindung der Formel I) und der
Maus (Verbindung der Formel II) sind im Folgenden gegeneinander
aufgereiht, um den hohen Grad von Sequenzidentität zu illustrieren.
-
-
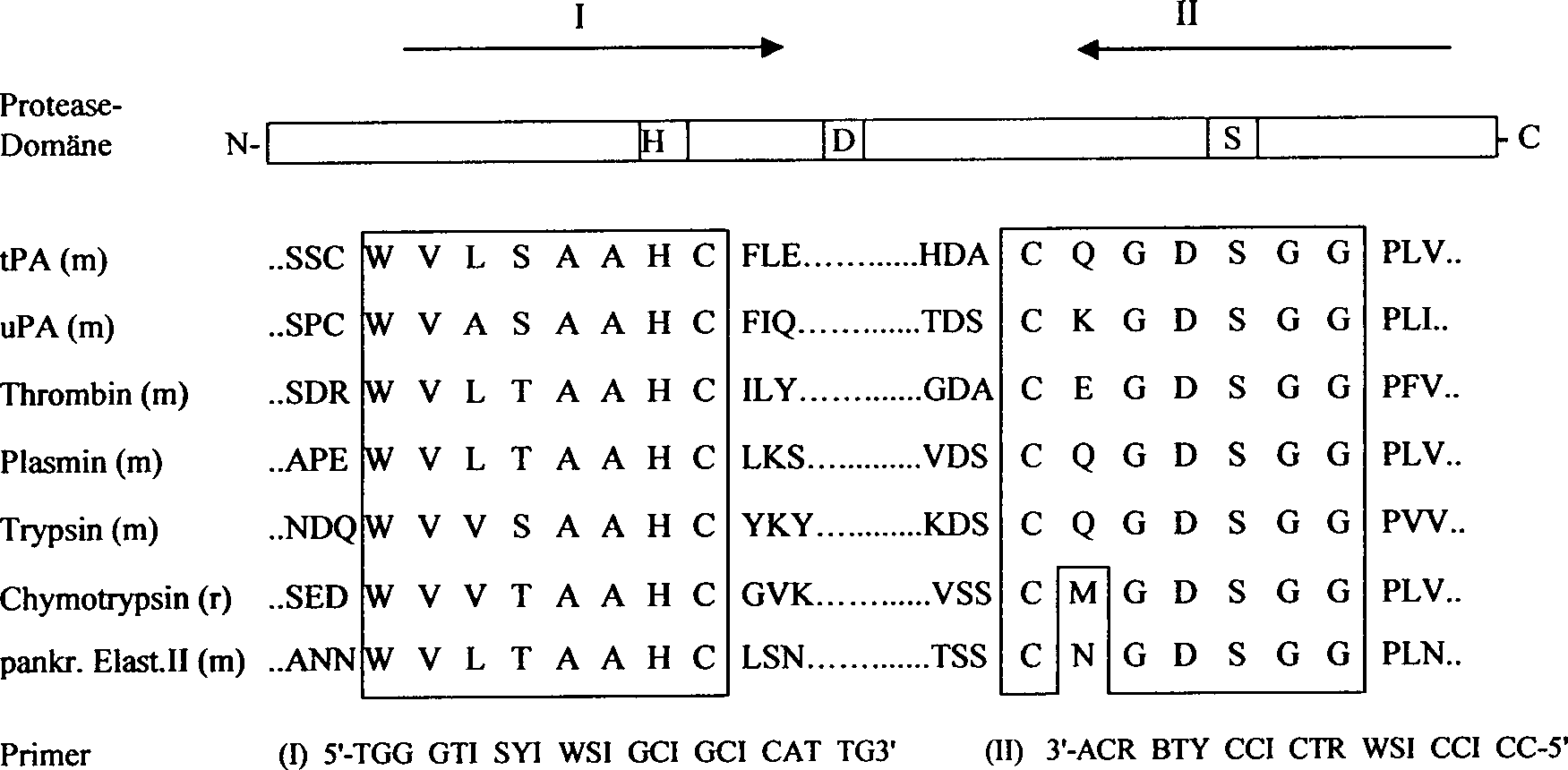
Von
258 Aminosäuresequenzpositionen,
welche in den Vergleich einbezogenen worden sind, sind 233 Aminosäuren in
beiden Verbindungen identisch (obere Sequenz: Verbindung der Formel
I; untere Sequenz: Verbindung der Formel II; identische Aminosäuren sind
mit senkrechten Strichen gekennzeichnet).
-
Die
erfindungsgemässen
Neurotrypsine sind verglichen mit den bekannten Serinproteasen einzigartig, weil
sie gemäss
gegenwärtigen
Erkenntnissen in ausgeprägtem
Masse von Nervenzellen exprimiert werden. Ein anderes Organ mit
starker Expression von Neurotrypsin ist die Lunge (siehe Gschwend
et al., Mol. Cell. Neurosci. 9, Seiten 207–219, 1997).
-
Die
den Strukturen der Verbindungen der Formeln I oder II am stärksten gleichenden
Proteine sind Serinproteasen, wie etwa Gewebe-Plasminogenaktivator
(tPA), Urokinase-Typ-Plasminogenaktivator (uPA), Plasmin, Trypsin,
Apolipoprotein (a), Coagulation-Factor XI, Neuropsin, und Acrosin.
-
Im
erwachsenen Gehirn werden die erfindungsgemässen Verbindungen vorwiegend
in der Grosshirnrinde, dem Hippocampus, und der Amygdala exprimiert.
-
Im
erwachsenen Hirnstamm und Rückenmark
werden die erfindungsgemässen
Verbindungen vorwiegend in den motorischen Nervenzellen exprimiert.
Eine etwas schwächere
Expression ist in den Nervenzellen der oberflächlichen Schichten des Hinterhorns
des Rückenmarks
zu finden.
-
Im
erwachsenen peripheren Nervensystem werden die erfindungsgemässen Verbindungen
in einer Subpopulation der Spinalganglienneurone exprimiert.
-
Die
erfindungsgemässen
Verbindungen wurden im Rahmen einer Studie gefunden, welche zum
Ziel hatte, Trypsin-ähnliche
Serinproteasen im Nervensystem aufzuspüren.
-
Die
erste Verbindung, die gefunden und charakterisiert wurde, war die
Verbindung der Formel II (siehe Gschwend et al., Mol. Cell. Neurosci.,
9, Seiten 207– 219,
1997).
-
Durch
ein "Alignment" der Proteasedomänen von
7 bekannten Serinproteasen (Gewebe-Typ Plasminogenaktivator, Urokinase-Typ
Plasminogenaktivator, Thrombin, Plasmin, Trypsin, Chymotrypsin und
pankreatische Elastase) in der Nähe
des Histidins und des Serins der katalytischen Triade der aktiven
Stelle wurden die Sequenzen von so genannten "Primer-Oligonukleotiden" für die Polymerasen-Kettenreaktion ermittelt.
-
Die
Primer-Oligonukleotiden wurden in einer Polymerasen-Kettenreaktion
(PCR) zusammen mit ss-cDNA aus Total-RNA aus Gehirnen von 10 Tage
alten Mäusen
eingesetzt und führten
zur Amplifizierung eines cDNA-Fragments mit einer Länge von
ungefähr
500 Basenpaaren.
-
Dieses
cDNA-Fragment wurde erfolgreich zur Isolierung von weiteren cDNA-Fragmenten mittels
der Durchsuche von im Handel erhältlichen
cDNA-Bibliotheken eingesetzt. Zusammen erstreckten sich die isolierten
cDNA-Fragmente über
die volle Länge
des kodierenden Teils der Verbindung der Formel II.
-
Durch
herkömmliche
DNA-Sequenzierung wurden die vollständige Nukleotidsequenz und
die davon abgeleitete Aminosäuresequenz
erhalten.
-
Die
Verbindung der Formel I wurde aufgrund ihrer ausgeprägten Ähnlichkeit
mit der Verbindung der Formel II kloniert.
-
Die
eingesetzten Primer-Oligonukleotide wurden gemäss der bekannten Sequenz der
Verbindung der Formel II synthetisiert.
-
Die
Klonierung der Verbindung der Formel I wurde mittels zweier im Handel
erhältlicher
cDNA-Bibliotheken aus fötalem
menschlichem Gehirn durchgeführt.
-
Diese
Art der Klonierung kann auch zur Isolierung der homologen Verbindung
anderer Spezies, wie Ratte, Kaninchen, Meerschweinchen, Rind, Schaf,
Schwein, Primaten, Vögel,
Zebrafisch (Brachydanio rerio), Drosophila melanogaster, Caenorhabditis
elegans etc., verwendet werden.
-
Die
kodierenden Nukleotidsequenzen können
eingesetzt werden zur Erzeugung von Proteinen mit den kodierten
Aminosäuresequenzen
der Verbindungen der Formeln I oder II. Ein in unserem Labor praktiziertes Verfahren
erlaubt die Produktion von rekombinanten Proteinen in Myelomazellen
als Fusionsproteine mit einer Immunoglobulin-Domäne (konstante Domäne der Leichten-Kette-Kappa).
Das Konstruktionsprinzip ist im Detail beschrieben durch Rader et
al. (Rader et al., Eur. J. Biochem. 215, Seiten 133–141, 1993).
Das so von den Myelomazellen synthetisierte Fusionsprotein wurde
durch Immunoaffinitätschromatographie
mittels eines monoklonalen Antikörpers
gegen die Ig-Domäne
der Leichten-Kette-Kappa isoliert. Mit der gleichen Expressionsmethode
kann auch das native Protein einer Verbindung, ausgehend von der
kodierenden Sequenz, produziert werden.
-
Die
kodierenden Sequenzen der Verbindungen der Formeln I oder II können als
Ausgangsverbindungen dienen für
das Aufspüren
und die Isolierung von Allelen der Verbindungen der Formeln I oder
II. Sowohl die Polymerasen-Kettenreaktion als auch die Nukleinsäure-Hybridisierung
können
zu diesem Zweck eingesetzt werden.
-
Die
kodierenden Sequenzen der Verbindungen der Formeln I oder II können als
Ausgangsverbindungen dienen für
so genannte "site-directed
mutagenesis", um
Nukleotidsequenzen zu generieren, welche die durch die Verbindungen
der Formeln I oder II kodierten Proteine, oder Teile davon, kodieren,
aber deren Nukleotidsequenz im Bezug auf die Verbindungen der Formeln
I oder II degeneriert sind, bedingt durch die Verwendung alternativer
Codons.
-
Die
kodierenden Sequenzen der Verbindungen der Formeln I oder II können verwendet
werden als Ausgangsverbindungen für die Herstellung von Sequenzvarianten
durch so genannte "site-directed
mutagenesis".
-
Beste Arten zur Ausführung der
Erfindung (Beispiele)
-
cDNA-Klonierung der Verbindung
der Formel II (Neurotrypsin der Maus)
-
Totale
RNA wurde aus dem Gehirn von 10 Tage alten Mäusen (ICR-ZUR) gemäss der Methode
von Chomczynski und Sacchi (1987) isoliert. Die Herstellung von
einzelsträngiger
cDNA erfolgte unter Benützung von
Oligo(dT)-Primer" und
einer RNA-abhängigen
DNA-Polymerase (Superscript RNase H–-Reverse
Transcriptase; Gibco-BRL, Gaithersburg, MD) gemäss den Instruktionen des Herstellers.
Für die
Durchführung
der Polymerasen-Kettenreaktion wurden ein vorwärts-gerichteter "Primer", gemäss der Aminosäuresequenz
der Region des konservierten Histidins der katalytischen Triade,
und ein rückwärts-gerichteter "Primer", gemäss der Aminosäuresequenz
der Region des konservierten Serins der katalytischen Triade der
Serinproteasen, synthetisiert. Die Aminosäuresequenzen, welche für die Bestimmung
der Oligonukleotid-„Primer" verwendet wurden,
wurden von 7 bekannten Serinproteasen entnommen. Sie sind im Folgenden
dargestellt.
-
-
Die
Protease-Domänen
von 7 bekannten Serinproteasen (Gewebe-Typ-Plasminogenaktivator, Urokinase-typ
Plasminogenaktivator, Thrombin, Plasmin, Trypsin, Chymotrypsin und
pankreatische Elastase) wurden im Bereich des konservierten Histidins
und Serins der katalytischen Triade der aktiven Stelle aufgereiht. Die
in diesen Regionen konservierten Aminosäuren wurden als Basis für die Bestimmung
der degenerierten "Primer" benutzt. Die Primersequenzen
sind nach der Empfehlung der IUB-Nomenklatur (Nomenclature Committee,
1985) angegeben.
-
Die
in der PCR eingesetzten Primer trugen zur Erleichterung einer späteren Klonierung
die Restriktionsstellen für
EcoRI und BamHI an ihren 5'-Enden.
-
Folgende
Primer wurden eingesetzt:
-
In
Leserichtung (sense primers):
5'-GGGGAATTCTGGGTI(C/G)(T/C)I(T/A)(G/C)IGCIGCICA(T/C)TG-3'
-
In
Gegenrichtung (antisense primers):
5'-GGGGGATCCCCICCI(G/C)(A/T)(A/G)TCICC(C/T)T(G/C/T)(G/A)CA-3'.
-
Die
Polymerasen-Kettenreaktion wurde unter Standard-Bedingungen mittels
der DNA-Polymerase AmpliTaq
(Perkin Elmer) gemäss
den Empfehlungen des Produzenten durchgeführt. Das folgende PCR-Profil wurde
eingesetzt: 93°C
für 3 Minuten,
gefolgt von 35 Zyklen von 93°C
für 1 Minute,
48°C für 2 Minuten
und 72°C
für 2 Minuten.
Im Anschluss an den letzten Zyklus wurde die Inkubation bei 72°C während weiteren
10 Minuten fortgesetzt.
-
Die
amplifizierten Fragmente hatten eine ungefähre Länge von 500 Basenpaaren. Sie
wurden mit EcoRI und BamHI geschnitten und in einen Bluescript-Vektor
eingefügt
(Bluescript SK(–),
Stratagene). Die resultierenden Klone wurden durch DNA-Sequenzbestimmung
mittels der Didesoxy-Kettenterminations-Methode (Sanger et al.,
Proc. Natl. Acad. Sci. USA 77, Seiten 2163–2167, 1977) auf einem automatisierten
DNA-Sequenziergerät
(LI-COR, Modell 4000L; Lincoln, NE) unter Benützung eines kommerziellen Sequenzierkits
(SequiTherm long-read cycle sequencing kit-LC; Epicentre Technologies,
Madison, WI) analysiert. Die Analyse führte zu einer Sequenz von 474
Basenpaaren der katalytischen Region der Serinprotease-Domäne der Verbindung
der Formel II.
-
Das
474 Basenpaar lange PCR-Fragment wurde zum Absuchen einer Oligo(dT)-"primed" Uni-ZAP-XR-cDNA-Bibliothek aus dem
Gehirn von 20 Tage alten Mäusen
(Stratagene; Cat. No. 937 319) eingesetzt. Es wurden 3 × 106 Lambda-Plaques mittels des radioaktiv markierten
PCR-Fragments als Sonde unter hochstringenten Bedingungen (Sambrook
et al., Molecular Cloning: A Laboratory Manual, Cold Spring Harbor Laboratory
Press, 1989) abgesucht und es wurden 24 positive Klone gefunden.
-
Aus
den positiven Lambda-Uni-ZAP-XR-Phagemid-Klonen wurde das entsprechende
Bluescript-Plasmid nach der Standardmethode gemäss den Empfehlungen des Herstellers
(Stratagene) durch in vivo-Exzision herausgeschnitten. Um die Länge der
eingefügten
Fragmente zu bestimmen, wurden die entsprechenden Bluescript-Plasmid-Klone mit
SacI und KpnI verdaut. Die Klone, welche die längsten Fragmente enthielten, wurden
mittels DNA-Sequenzierung (wie oben beschrieben) analysiert, und
für die
anschliessende Daten-Auswertung wurde die GCG-Software (Version
8.1, Unix; Silicon Graphics, Inc.) verwendet.
-
Da
keiner der Klone die kodierende Sequenz in voller Länge enthielt,
wurde eine zweite cDNA-Bibliothek abgesucht. Die in dieser Absuche
eingesetzte Bibliothek war eine Oligo(dT)- und "Random-Primed" cDNA Bibliothek in einem Lambda-Phagen
(Lambda gt10), welche auf mRNA aus 15 Tage alten Maus-Embryonen basierte
(oligo(dT)- and random-primed Lambda gt10 cDNA library; Clontech,
Palo Alto, CA; Kat. No. ML 3002a). Als Sonde wurde ein radioaktiv
markiertes DNA-Fragment (AvaI/AatII) vom 5'-Ende des längsten Klones der ersten Suche
eingesetzt, und es wurden ungefähr
2 × 106 Plaques abgesucht. Diese Absuche ergab 14
positive Klone. Die cDNA-Fragmente wurden mittels EcoRI herausgeschnitten
und in den Bluescript-Vektor (KS(+);
Stratagene) kloniert. Die Sequenzanalyse wurde wie oben beschrieben
ausgeführt.
-
Man
erhielt so die Nukleotidsequenz über
die volle Länge
der cDNA von 2361 resp. 2376 Basenpaaren der Verbindung der Formel
II. Mit dem beschriebenen Verfahren der PCR-Klonierung ist es möglich, auch
Varianten-Formen der Verbindungen der Formeln I und II zu finden
und zu isolieren, beispielsweise deren Allele, oder deren Splice-Varianten.
Das beschriebene Verfahren des Absuchens einer cDNA-Bibliothek ermöglicht auch
das Auffinden und die Isolierung von Verbindungen, welche unter
stringenten Bedingungen an die kodierenden Sequenzen der Formeln
I und II hybridisieren.
-
Klonierung der cDNA der
Verbindung der Formel I (Neurotrycisin des Menschen)
-
Die
Klonierung der cDNA der Verbindung der Formel I wurde auf der Grundlage
der Nukleotidsequenz der Verbindung der Formel II durchgeführt. Als
erster Schritt wurde ein Fragment der Verbindung der Formel I mittels
Polymerasenkettenreaktion (PCR) amplifiziert. Als Matrize wurde
die DNA verwendet, welche aus einer cDNA-Bibliothek vom Gehirn eines
menschlichen Fötus
(17.–18.
Schwangerschaftswoche) erhalten wurde, welche auf dem Markt erhältlich ist
(Oligo(dT)- and random-primed, human fetal brain cDNA library in
the Lambda ZAP II vector, Cat. No. 936206, Stratagene). Die synthetischen
PCR-Primer enthielten, zur Erleichterung der nachfolgenden Klonierung,
die Restriktionsstellen HindIII und XhoI am 5'-Ende.
-
In
Leserichtung (sense primers):
5'-GGGAAGCTTGGICA(A/G)TGGGGIACI(A/G)TITG(C/T)GA(C/T)-3'
-
In
Gegenrichtung (antisense primer):
5'-GGGCTCGAGCCCCAICCTGTTATGTAAIAGTTG-3'
-
Die
PCR wurde unter Standard-Bedingungen mittels der DNA-Polymerase
Amplitaq (Perkin Elmer) gemäss
den Empfehlungen des Produzenten durchgeführt. Das entstandene Fragment
von 1116 Basenpaaren wurde in den Bluescript-Vektor (Bluescript
SK(–),
Stratagene) eingefügt.
Ein 600 Basenpaare-langes HindIII/StuI-Fragment, entsprechend der 5'-Hälfte des
1116 Basenpaare-langen PCR-Fragments, wurde zum Absuchen einer Lambda-cDNA-Bibliothek
aus menschlichem fötalem
Gehirn (Human Fetal Brain 5'-STRETCH PLUS
cDNA library; Lambda gt10; Cat. No. HL3003a; Clontech) eingesetzt.
Es wurden 2 × 106 Lambda-Plaques mittels des radioaktiv markierten
PCR-Fragments unter hochstringenten Bedingungen (Sambrook et al.,
Molecular Cloning: A Laboratory Manual, Cold Spring Harbor Laboratory
Press, 1989) abgesucht, und es wurden 23 positive Klone gefunden
und isoliert.
-
Aus
den positiven Lambda-gt10-Klonen wurden die entsprechenden cDNA-Fragmente mit EcoRI
herausgeschnitten und in einen Bluescript-Vektor (Bluescript KS(+),
Stratagene) eingefügt.
Die Sequenzierung erfolgte mittels der Didesoxy-Kettenterminations-Methode (Sanger et
al., Proc. Natl. Acad. Sci. USA 77, Seiten 2163–2167, 1977), unter Verwendung
eines kommerziellen Sequenzierkits (Sequi Therm long-read cycle
sequencing kit-LC; Epicentre Technologies, Madison, WI) und Bluescript-spezifischen
Primern.
-
In
einer alternativen Sequenzier-Strategie wurden die cDNA-Fragmente
der positiven Lambda-gt10-Klone, unter Verwendung Lambda-spezifischer
Primer, mittels PCR amplifiziert. Die Sequenzierung wurde wie oben
beschrieben durchgeführt.
-
Die
computerisierte Analyse der Sequenzen wurde mittels des Programmpakets
GCG (Version 8.1, Unix; Silicon Graphics Inc.) durchgeführt.
-
Man
erhielt so die Nukleotidsequenz über
die volle Länge
der cDNA von 3350 Basenpaaren. Mit dem beschriebenen Verfahren der
PCR-Klonierung ist es möglich,
auch Varianten-Formen der Verbindungen der Formeln I oder II zu
finden und zu isolieren, beispielsweise deren Allele, oder deren
Splice-Varianten. Das beschriebene Verfahren des Absuchens einer
cDNA-Bibliothek ermöglicht
auch das Auffinden und die Isolierung von Verbindungen, welche unter
stringenten Bedingungen mit den codierenden Sequenzen der Formeln
I oder II hybridisieren.
-
Visualisierung der codierten
Sequenzen der Verbindungen I oder II mittels Antikörpern
-
Das
mehr als 60 Aminosäuren
lange Prolin-reiche, basische Segment am Aminoterminus der codierten
Sequenz der Verbindungen der Formeln I oder II eignet sich gut für die Herstellung
von Antikörpern
mittels der Synthese von Peptiden und deren Einsatz zur Immunisierung.
Wir haben aus dem Prolin-reichen, basischen Segment am Aminoterminus
der kodierten Sequenz der Verbindung der Formel II zwei Peptidsequenzen
mit einer Länge
von 19 und 13 Aminosäuren
zur Erzeugung von Antikörpern
ausgewählt.
Die Peptide hatten die folgenden Sequenzen:
Peptid 1: H2N-SRS PLH RPH PSP PRS QX-CONH2
Peptid
2: H2N-LPS SRR PPR TPR F-COOH
-
Die
beiden Peptide wurden chemisch synthetisiert, an eine makromolekulare
Trägersubstanz
(Keyhole Limpet Hemacyanin) gekoppelt, und zur Immunisierung in
2 Kaninchen injiziert. Die erzeugten Antiseren wiesen einen hohen
Antikörper-Titer
auf und konnten erfolgreich sowohl zur Identifizierung von nativem
Neurotrypsin aus Gehirnextrakt der Maus als auch zur Identifizierung
von rekombinantem Neurotrypsin verwendet werden. Das angewandte
Verfahren zur Erzeugung von Antikörpern kann auch zur Erzeugung
von Antikörpern gegen
die kodierte Sequenz der Verbindung der Formel I angewendet werden.
-
Die
resultierenden Antikörper
gegen die Teilsequenzen der kodierten Sequenzen der Formeln I oder II
können
zur Aufspürung
und zur Isolierung von Varianten-Formen der Verbindungen der Formeln
I oder II, wie beispielsweise Allele oder Splice-Varianten, eingesetzt
werden. Solche Antikörper
können
auch verwendet werden für
die Auffindung und Isolierung von gentechnisch erzeugten Varianten
der Verbindungen der Formeln I oder II.
-
Reinigung der kodierten
Sequenzen der Verbindungen der Formeln I oder II
-
Neben
konventionellen chromatographischen Methoden, wie beispielsweise
Ionenaustauscher-Chromatographie, kann die Reinigung der kodierten
Sequenzen der Verbindungen der Formeln I oder II, auch erreicht
werden unter der Verwendung von zwei affinitätschromatographischen Reinigungsverfahren.
Eine affinitätschromatographische
Reinigungsprozedur basiert auf der Verfügbarkeit von Antikörpern. Durch
Kopplung der Antikörper
an eine chromatographische Matrix resultiert ein Reinigungsverfahren,
in welchem ein sehr hoher Grad an Reinheit der entsprechenden Verbindung
in einem Schritt erzielt werden kann.
-
Ein
anderes wichtiges Merkmal, welches für die Reinigung der kodierten
Sequenzen der Verbindungen der Formeln I und II verwendet werden
kann, ist das Prolin-reiche, basische Segment am Aminoterminus. Es
ist zu erwarten, dass, aufgrund der hohen Dichte an positiven Ladungen,
dieses Segment die Bindung der kodierten Sequenzen der Verbindungen
der Formeln I oder II an Heparin und Heparin-ähnliche Affinitätsmatrices
vermittelt. Dieses Prinzip ermöglicht
auch die Isolierung, oder zumindest die Anreicherung, von Varianten-Formen
der kodierten Sequenzen der Formeln I oder II, beispielsweise deren
Allele oder Splice-Varianten. Gleichermassen kann die Heparin-Affinitätschromatographie
auch zur Isolierung, oder zumindest zur Anreicherung, von spezieshomologen
Proteinen der Verbindungen der Formeln I oder II eingesetzt werden.
-
Industrielle
Anwendbarkeit
-
Die
kodierenden Sequenzen der Verbindungen der Formeln I und II können verwendet
werden für
die Herstellung der kodierten Proteine, oder Teilen davon, der Formeln
I und II. Die Herstellung der kodierten Proteine kann in prokaryotischen
oder eukaryotischen Expressionssystemen erzielt werden.
-
Das
Gen-Expressionsmuster der erfindungsgemässen Verbindungen im Gehirn
ist äusserst
interessant, weil diese Moleküle
im adulten Nervensystem vor allem in Nervenzellen derjenigen Regionen
exprimiert werden, denen eine wichtige Rolle bei Lern- und Gedächtnisfunktionen
zugeschrieben wird. Zusammen mit der kürzlich gefundenen Evidenz für eine Rolle
von extrazellulären
Proteasen bei der neuralen Plastizität, lässt das Expressionsmuster vermuten,
dass die proteolytische Wirkung von Neurotrypsin eine Rolle innehat
bei strukturellen Reorganisationen im Zusammenhang mit Lern- und
Gedächtnis-Operationen,
zum Beispiel Operationen, welche an der Verarbeitung und Speicherung
von erlernten Verhaltensweisen, erlernten Gefühlen oder von Gedächtnisinhalten
beteiligt sind. Die erfindungsgemässen Verbindungen können deshalb
Ziele für pharmazeutische
Interventionen bei Funktionsstörungen
des Gehirns sein.
-
Das
Gen-Expressionsmuster der erfindungsgemässen Verbindungen in der Grosshirnrinde
(vor allem Schichten V und VI) ist äusserst interessant, weil eine
Reduktion der zellulären
Differenzierung in der Grosshirnrinde in Assoziation mit Schizophrenie
gefunden wurde. Die erfindungsgemässen Verbindungen können deshalb
Ziele für
pharmazeutische Interventionen bei Schizophrenie und verwandten
psychiatrischen Krankheiten sein.
-
Es
ist gefunden worden, dass die kodierenden Sequenzen der erfindungsgemässen Verbindungen
in Neuronen erhöht
sind, welche an das beschädigte
Gewebe eines fokalen ischämischen
Hirnschlags angrenzen, was darauf hinweist, dass die erfindungsgemässen Verbindungen
eine Rolle in der Gewebereaktion in verletztem zerebralem Gewebe
spielen. Die erfindungsgemässen
Verbindungen können
deshalb Ziele für pharmazeutische
Interventionen nach einem ischämischen
Hirnschlag und anderen Formen von Beschädigungen von neuralem Gewebe
sein.
-
Vom
Gewebe-Typ Plasminogenaktivator, eine Serinprotease, welche mit
den erfindungsgemässen Verbindungen
verwandt ist, wurde kürzlich
gefunden, dass er in Excitotoxizitäts-vermittelten neuronalen
Zelltod involviert ist. Eine ähnliche
Funktion ist denkbar für
die erfindungsgemässen
Verbindungen und, folglich, stellen die erfindungsgemässen Verbindungen
ein mögliches
Ziel für
pharmakologische Interventionen bei Krankheiten dar, in welchen
der Zelltod auftritt.
-
Das
Gen-Expressionsmuster der erfindungsgemässen Verbindungen im Rückenmark
und in den sensorische Ganglien ist interessant, weil diese Moleküle im adulten
Nervensystem in Neuronen derjenigen Gehirnregionen exprimiert werden,
denen eine Rolle bei der Verarbeitung von Schmerz, sowie bei der
Entstehung pathologischer Schmerzzustände, zugeschrieben wird. Die
erfindungsgemässen
Verbindungen können
deshalb Ziele für
pharmazeutische Interventionen bei pathologischem Schmerz sein.
-
Im
folgenden Teil werden Angaben bezüglich der Verbindungen der
Formeln I oder II gemacht:
-
(1)
ANGABEN ZUR VERBINDUNG DER FORMEL I (Neurotrypsin des Menschen)
-
-
-
-
-
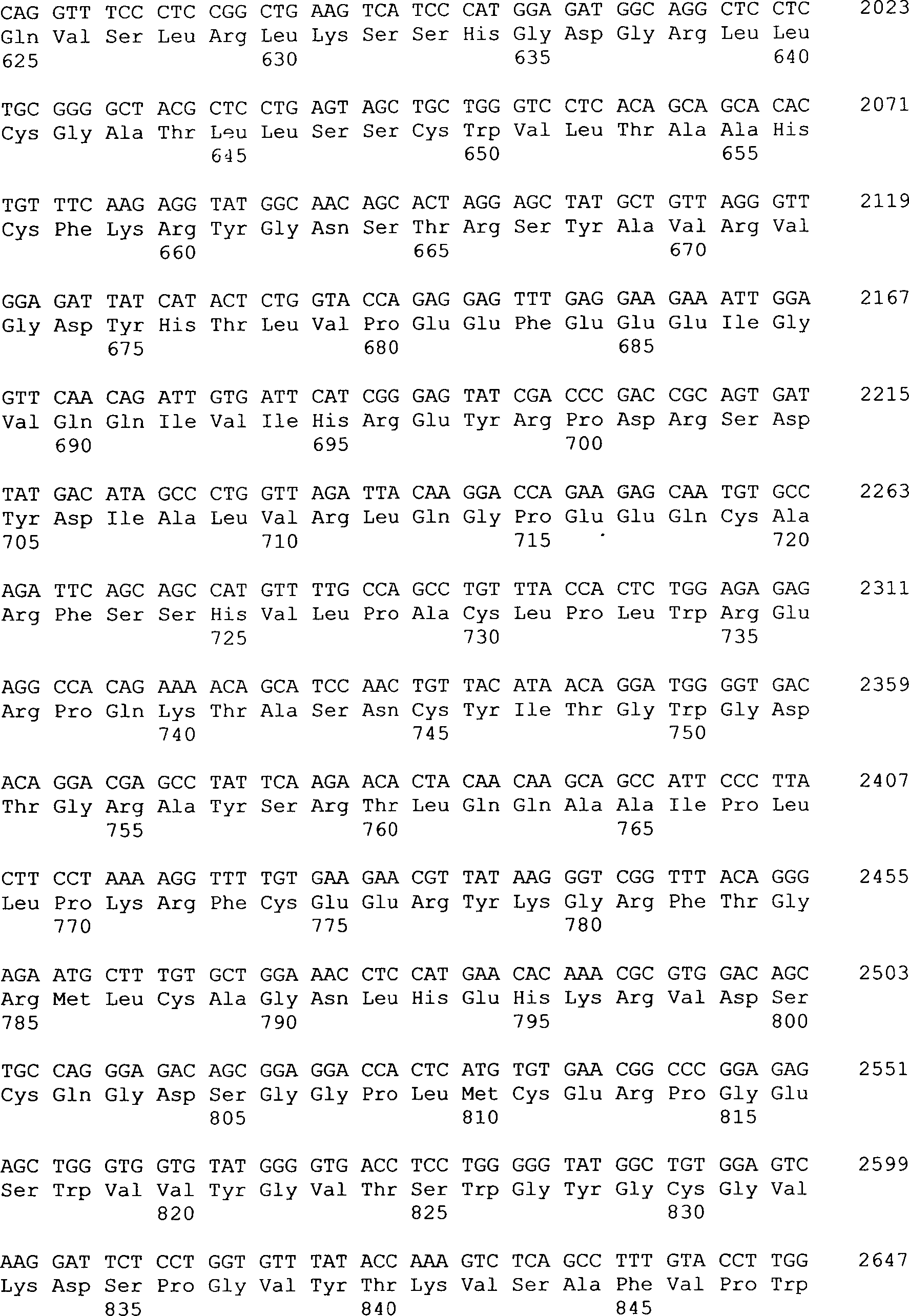
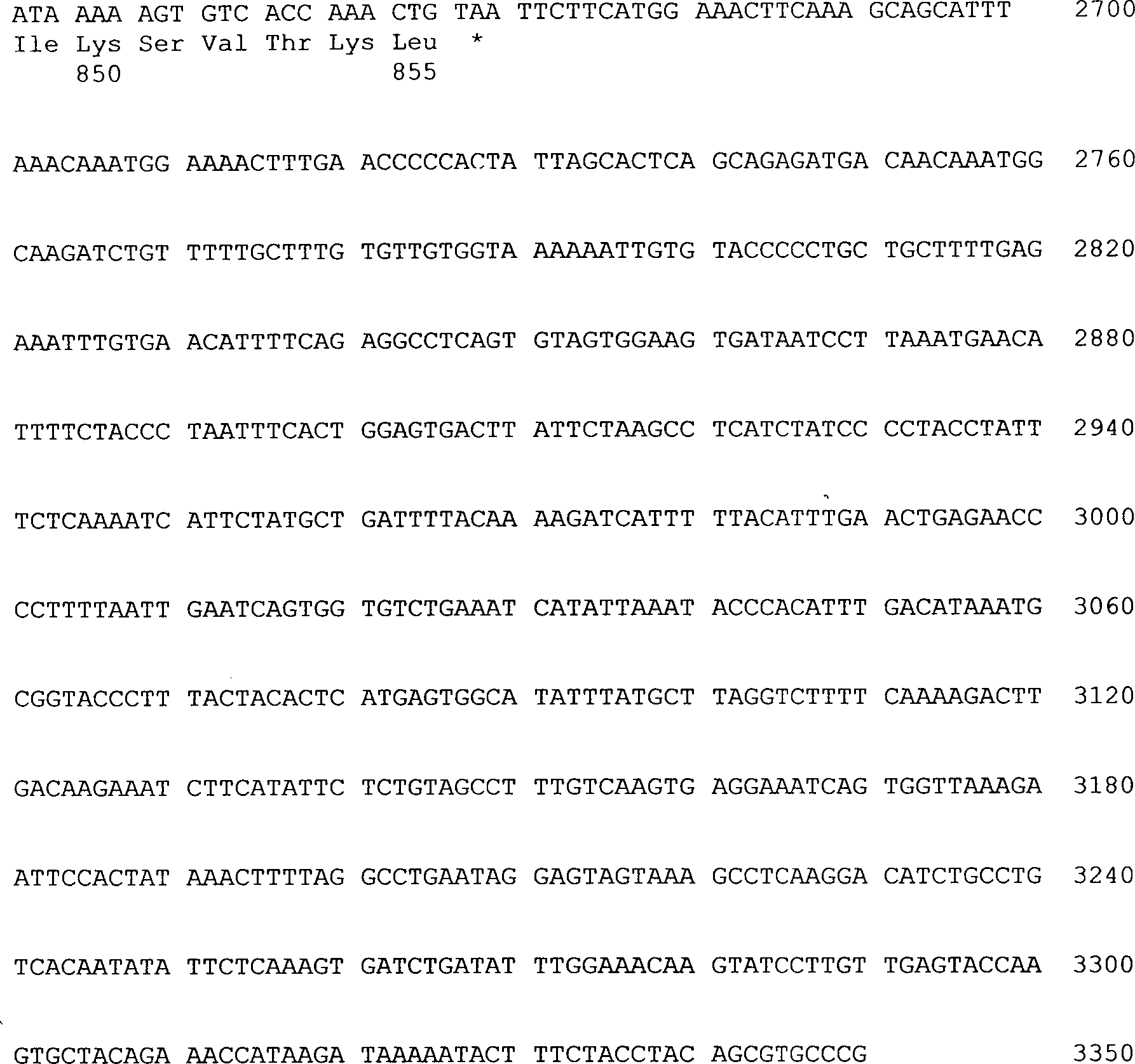
Verbindung
der Formel I (Neurotrypsin des Menschen)
-
-
-
-
-
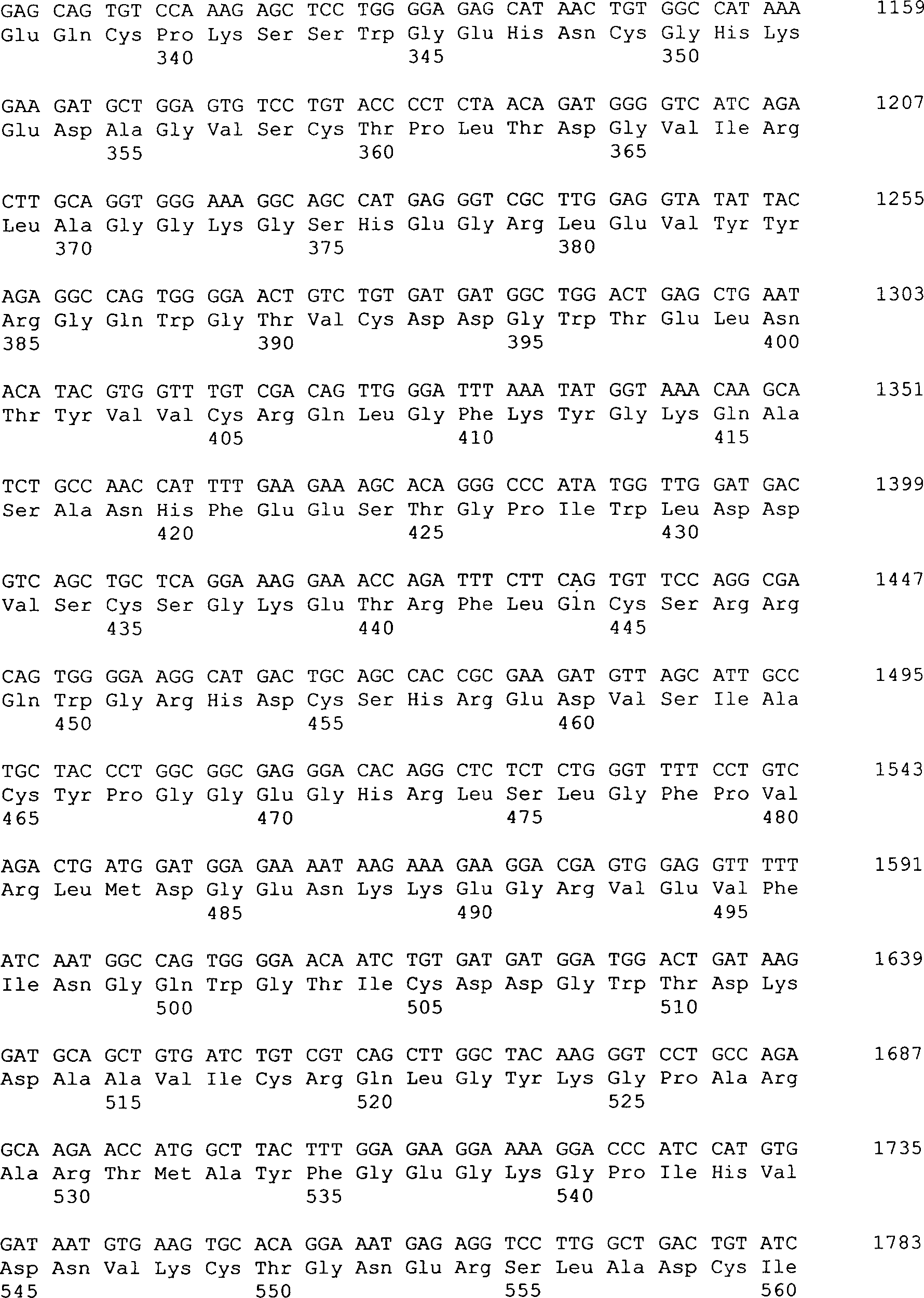
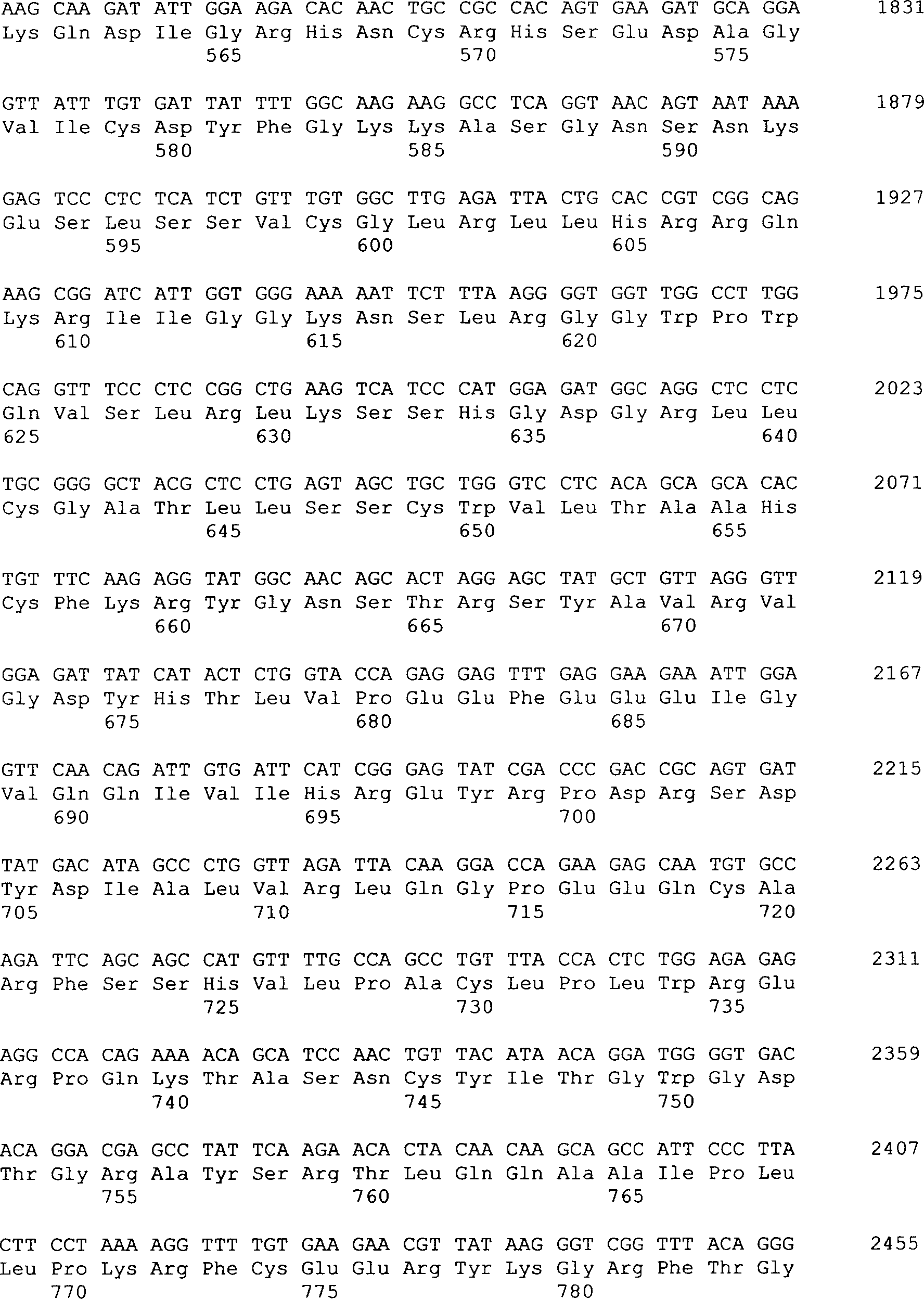
(1)
ANGABEN ZUR VERBINDUNG DER FORMEL II (Neurotrypsin der Maus)
-
-
-
-
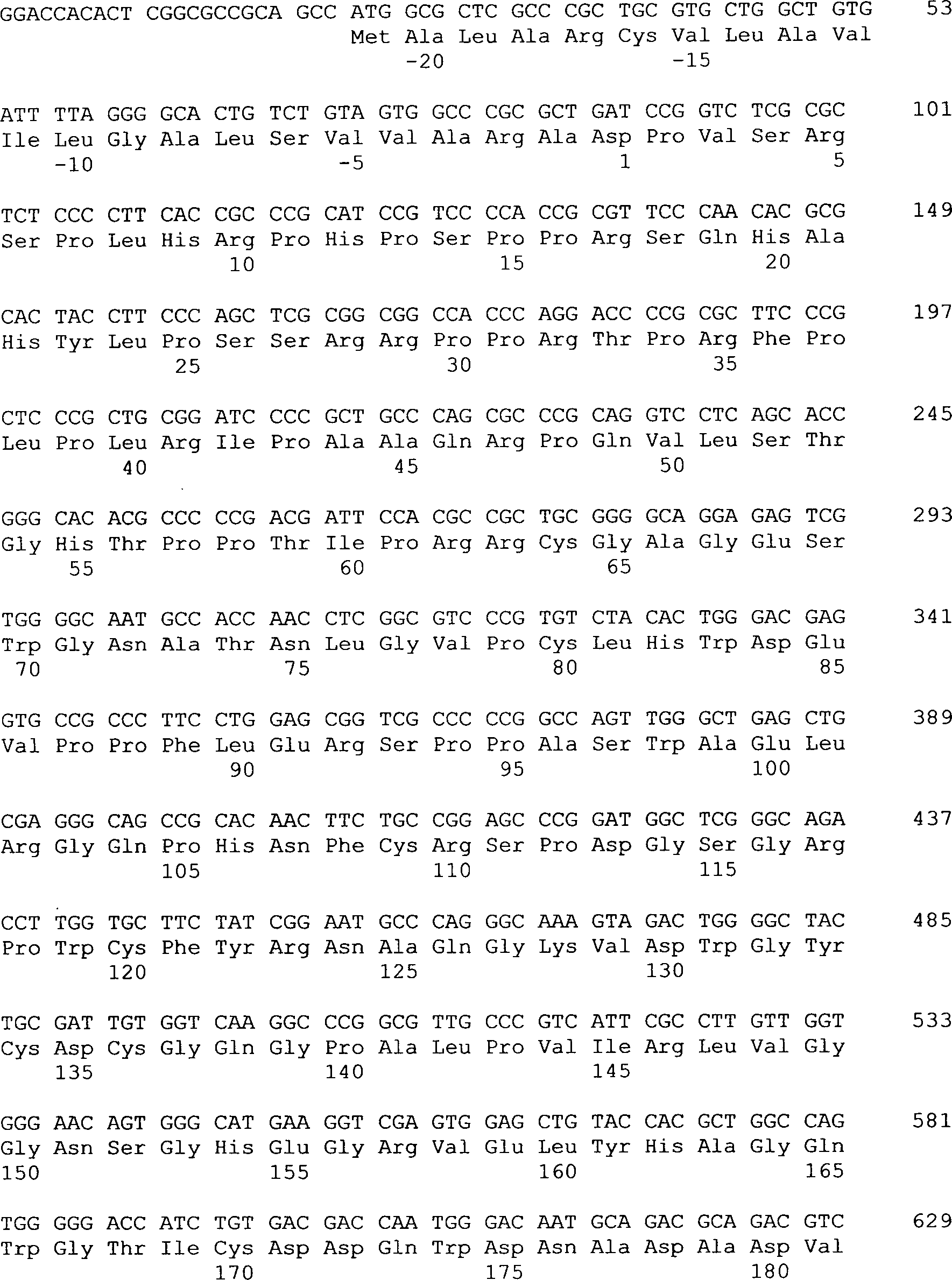
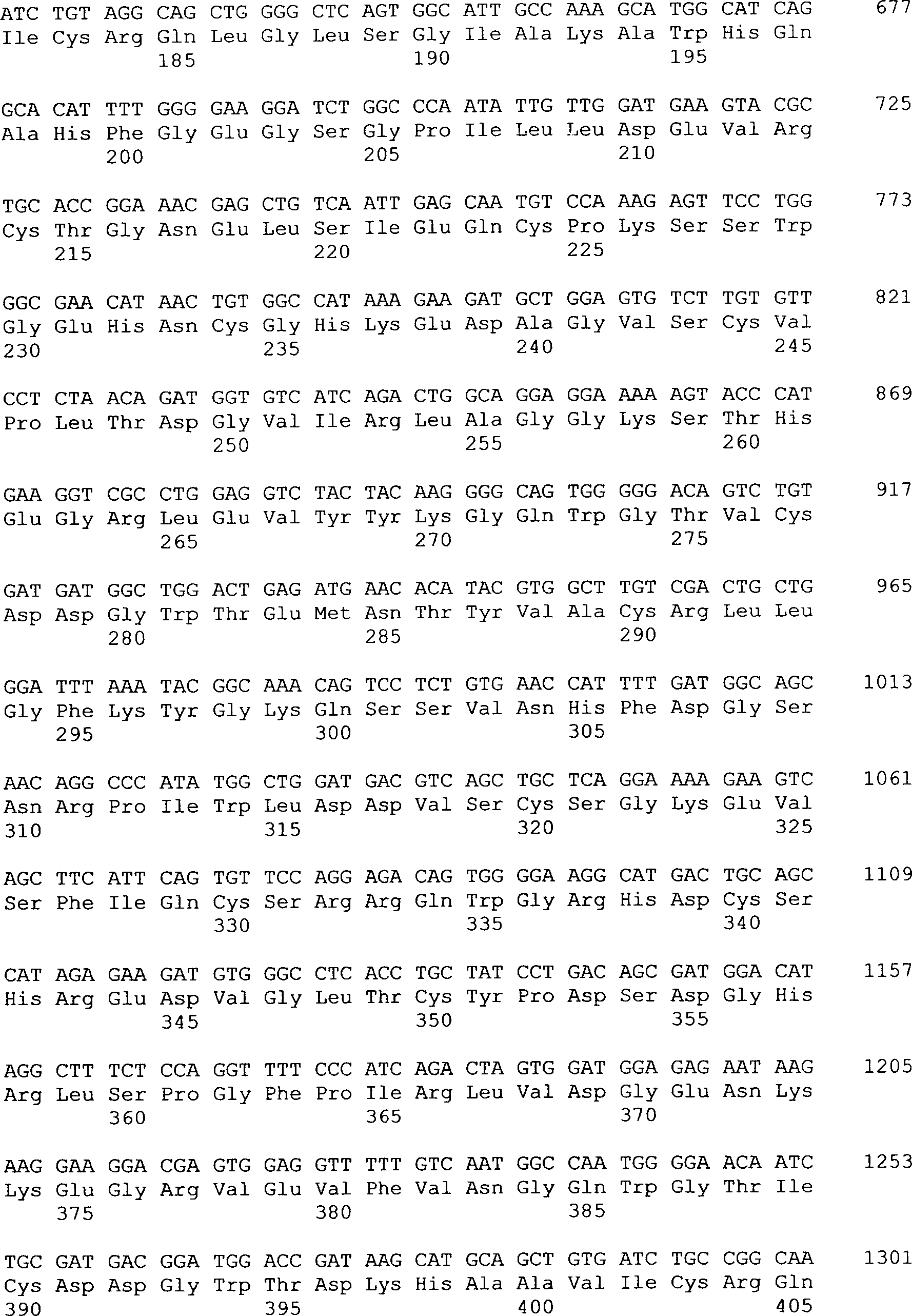
Verbindung
der Formel II (Neurotrypsin der Maus)
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-