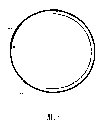稳定的腺病毒载体及其增殖方法
发明领域
本发明涉及医药学领域,尤其涉及重组腺病毒载体及其应用。
发明背景
人腺病毒是大小为60-90nm的无包膜二十面体颗粒。迄今为止已经鉴别了51种血清型,基于血细胞凝集性质及序列同源性将它们再分为6个亚群(Francki et al.1991)。基因组长度为34-36kb,其两侧是末端反向重复序列(ITR)。病毒感染周期分为早期和晚期阶段。在早期阶段(感染后6-8小时),病毒被脱壳且基因组转运进细胞核,之后早期基因区E1-E4成为转录活性的。
早期区-1(E1)含有称为E1A和E1B的两个转录区。E1A区编码参与宿主细胞周期修饰及其它病毒转录区激活的两个主要蛋白质(参见Russell,2000)。E1B区编码两个主要蛋白质,19K和55K,其通过不同途径阻止得自E1A蛋白活性的凋亡的诱导(Rao et al.,1992;Yew and Berk,1992;参见Shenk,1996)。另外,E1B-55K蛋白是在晚期阶段选择性病毒mRNA转运及抑制宿主蛋白质表达所需要的(Pilder et al.,1986)。早期区-2(E2)也分为E2A和E2B区,其一起编码三种蛋白质,DNA结合蛋白、病毒聚合酶和终端前蛋白质,这3种蛋白质均参与病毒基因组的复制(参见van der Vliet,1995所述)。E3区不是体外复制所必需的,但编码一些破坏宿主的病毒感染防御机制的蛋白质(参见Horwitz,2001所述)。E4区编码至少6种蛋白质,所述蛋白质参与与病毒mRNA剪接和转运、宿主细胞mRNA转运、病毒和细胞转录及转化相关的一些独特功能(参见Leppard,1997所述)。
病毒壳体的形成及病毒基因组的包装所需的晚期蛋白均产生自主要晚期转录单位(MLTU),其在复制开始后成为充分活性的。差别剪接和聚腺苷酸化的复杂过程产生15个以上共有一个三联前导序列的mRNA物种。早期蛋白E1B 55K和E4 Orf 3及Orf6在调节晚期病毒mRNA加工和从核中的转运中起关键作用(参见Leppard,1998所述)。
新形成的病毒基因组在先成的壳体中的包装由至少两种腺病毒蛋白质即晚期蛋白52/55K和中期蛋白IVa2介导,这通过与位于基因组左侧末端的包装序列的相互作用而进行(Grable and Hearing,1990;Gustin and Imperiale,1998;Zang et al.,2001)。另一种中期蛋白pIX是壳体的一部分而且已知其稳定六邻体一六邻体之间的相互作用(Furcinitti et al.,1989)。另外,已经将pIX描述为反式激活含有TATA的启动子如E1A启动子和MLP(Lutz et al.,1997)。
由于对病毒生物学的广泛了解及在进入细胞之后的高效核输送,因此腺病毒已经成为将基因输送进人体细胞的常用工具。另外,腺病毒载体是稳定的并可以相对简易地大规模生产。在大多数情况中,载体缺失至少E1区,这使所述载体产生复制缺陷。E1缺失的基于亚群C血清型Ad5或Ad2的载体的生产在E1互补细胞系如293(Graham etal.,1970),911(Fallaux et al.,1996)和PER.C6TM(Fallaux et al.,1998)中完成。正如美国专利5994128所揭示的,载体和细胞系需要仔细匹配以避免通过在所述细胞系和载体中腺病毒序列之间的同源重组而产生有复制能力的腺病毒。因此,PER.C6TM细胞及匹配的腺病毒载体提供了生产C群腺病毒载体的一种优选的系统(Fallaux et al.,1998)。E1序列的缺失提供了将外源基因导入病毒载体中的空间。由于可以掺入病毒体的Ad5基因组的最大大小限于大约wt长度的105%,因此E1缺失的病毒可以容纳大约4.8kb的外源DNA(Bett etal.,1993)。
缺少pIX的病毒体中最大包装能力降低至正常基因组长度的大约95%(Ghosh-Choudhury et al.,1987)。这最可能是由pIX-(pIX-minus)病毒体的稳定性降低所致。pIX-minus突变体Ad5中的缺乏可以由pIX在用于生产病毒的包装细胞系中的附加型表达而补足(Caravokyri etal.,1995)。
尽管血清型Ad5和Ad2最常用作基因转移载体,但其它血清型也可以具有优选的特性使其更适用作治疗或预防工具。例如亚群B病毒Ad35和Ad11与Ad5和Ad2病毒相比不易于被人血清中和(见WO00/70071所揭示)。
腺病毒转移载体的中和使体内转导效力降低。另外,发现与Ad5病毒相比,携带Ad35的尾丝的重组病毒对抗原呈递细如树突细胞的感染效力在体外显著增强(WO00/70071,WO02/24730)。因此,基于Ad35的载体组合了在人血清中低中和度的高度改良的感染效力,使这种载体适于进行免疫接种。
使用下文揭示的技术可以产生和增殖完全E1缺失的基于Ad35的载体。然而,对多种重组基于Ad35的载体进行仔细分析表明这种载体的稳定性较差,即与基于Ad5的载体相比可容纳较少量的外源DNA。本发明的手段和方法解决了这个问题。
另外,需要进一步揭示针对具有更广泛血清型用途的腺病毒的可利用的技术。现有的包装细胞系典型地包含衍生自腺病毒血清型5的E1编码的蛋白质。这种‘标准’包装细胞系例如是293、911和PER.C6TM。在这些标准包装细胞系上生产衍生自其它血清型的载体的尝试不是不成功就是很费力。根据所用的特殊血清型偶尔见到一些产品。然而,衍生自除了亚群C之外的腺病毒亚群的,由Ad5的E1转化和无限增殖化的细胞系上生产的重组腺病毒载体的产量很低。在Abrahamsen等(1997)的论文中,揭示了与缺乏Ad5的E4-orf6序列的293细胞相比,在包含衍生自腺病毒血清型5的E4-orf6的293细胞上观测到E1A缺失的腺病毒血清型7载体(亚群B)的改进的噬斑纯化。然而,由于在噬斑纯化的原种中观测到不希望的重组而遇到载体稳定性的问题。另一个问题是在生产期间野生型腺病毒污染问题。另外,就腺病毒的大规模生产而言,共同转染E4-orf6以获得足够高到可以应用的滴度是无用的。生长这种腺病毒的一个选则是提供具有稳定整合进互补/包装细胞系的基因组中的E4-orf6基因的细胞。这种细胞在本领域已经加以描述(例如WO96/22378所述)。这个系统的缺点是必须产生新的稳定细胞系,而且在产生稳定及正确的细胞之前必须进行许多次选择循环。这个过程费力又费时。通常地,可以确定来自除了血清型5(亚群C)之外的血清型的腺病毒如亚群B病毒的产生和增殖在Ad5互补细胞上难以进行。正如WO00/70071所揭示的,基于亚群B病毒Ad35的重组病毒可以通过含有Ad35早期区-1序列(Ad35-E1)的一表达构建体的共转染而产生。另外,仅缺失E1A序列而未缺失E1B序列的基于Ad35的病毒示出在PER.C6细胞上有效复制,提示Ad5的E1A蛋白能互补Ad35-E1A功能(WO02/40665)。此外,该实验示出缺少Ad35-E1B导致在Ad5互补细胞上产量较少。WO00/70071还揭示了生产E1缺失的非C群腺病毒载体的细胞系,通过进一步修饰能互补腺病毒血清型5的细胞系进行。WO00/70071进一步提示应确立携带Ad35-E1序列的新的细胞系以互补缺少E1区的重组腺病毒血清型35载体(见WO02/40665所述)。然而,正如上所述,如果针对特别需要而应用一种特异的血清型,不得不针对每一种特异的血清型确立一种新的细胞系,或者不得不修饰能互补腺病毒血清型5的可利用的细胞系以互补感兴趣的血清型。这对于应用本领域已知的确立并有效的方法,应用本领域可利用的确立细胞系而不经修饰应用这些细胞系生产所有其它的非Ad5血清型显然是有利的。
可以推断仍需要一种生产系统以生产与亚群C血清型不同的有用产量的腺病毒血清型。另外,仍需要包含常规包装细胞及稳定的并可以在这种包装细胞上增殖的重组亚群B腺病毒的合适的包装系统。
附图简述
图1:pWE.Ad35.pIX-rITRΔE3的图谱。
图2:在有和无E3区的Ad35E1缺失的病毒上产生的PCR片段的凝胶分析。P1=质粒对照;M=标记:1kb plus ladder(Invitrogen);mQ=H2O。基因组长度以kb标示。
图3A:用MEGalign软件(DNAstar)使用Clustal方法产生的多种腺病毒的近端pIX上游序列区的序列对比。序列来源在文中指明。方框内的是Ad5和Ad2中的Sp1位点和TATA框(见Babiss和Vales,1991所述)。
图3B:图3A中给出的序列的近端pIX区中推定的Sp1和TATA框的示意性对比。
图4:pAdApt535.Luc的图谱。
图5:pAdApt535的图谱。
图6A:在分离自Ad35.AdApt.Luc和Ad35.AdApt535.Luc病毒的DNA上产生的或者在对照质粒上产生的PCR片段的凝胶分析。M=标记(1kb plus ladder,Invitrogen)。使用两个特异性PCR扩增分析每个病毒制备物或对照质粒。
图6B:在Ad35.AdApt.LacZΔE3(35LacZ)和Ad35.AdApt535.LacZΔE3(535LacZ)病毒上或者在对照质粒上产生的PCR片段的凝胶分析。
图7:pBr.Ad35.ΔSM.AdApt.LacZ的图谱。
图8:E1B启动子和55K编码区中推定的启动子的示意图。
图9:可用于产生独特片段以鉴别推定的启动子的55K区域中限制位点的示意图。位置编号根据其在野生型Ad35中的位置而定。
图10:三种不同的亚群B血清型中E1A区和E1B/pIX区的polyA信号之间的区域的序列对比。
图11:质粒pΔMT.Orf6.Hygro(ECACC保藏号P02041226)的示意图。
图12:质粒pAd35.ΔMT.Orf6的示意图。
图13:pUC.35-5E4的示意图。
图14:产生pUC.35-5E4的克隆步骤。
图15:pBr.Ad35.PRn的示意图。
图16:pBr.Ad35.PR5E4(ECACC保藏号P02041229)的示意图。
图17:pWE.Ad35.pIX-rITR5E4的示意图。
图18:pCRscriptAmp.NFI-NcoIR的示意图。
图19:pCRscriptAmp.NcoIF-NR2的示意图。
图20:pCR.NF1-NR2的示意图。
图21:Ad5的E4-orf6(上方序列)与克隆进Ad35主链中的Ad5的E4-orf6(中间序列)及Ad35E4-orf6序列(下方序列)之间的序列对比,示出所述完整片段已经被置换。
图22:Ad5的E4-orf6/7(上方序列)与克隆进Ad35主链中部分Ad5E4-orf6/7(中间序列)及Ad35E4-orf6/7序列(下方序列)之间的序列对比,示出orf6/7序列是部分嵌合的,大约在第138位的赖氨酸(K)残基融合。
图23.pBr.Ad35.PR.5Orf6(ECACC保藏号P02041227)的示意图。
图24:pWE.Ad35.pIX-rITR.5Orf6的示意图。
图25:pBr.Ad35.PRnΔE3的示意图。
图26:pBr.Ad35.ΔE3.PR5E4的示意图。
图27:pBr.Ad35.ΔE3.PR5Orf6的示意图。
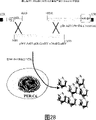
图28:在如PER.C6的细胞中通过双同源重组(double homologousrecombination)生产重组腺病毒颗粒的系统的示意图。
图29:pWE.Ad35.pIX-EcoRV的示意图。
图30:Ad35及Ad11pIX-cDNA序列和wtAd35序列的对比。
使用来自DNASTAR的SeqMan软件对比得自实施例18中所述的克隆的cDNA片段的序列。Ad35 cDNA序列衍生自分离自wtAd35或Ad35E1B+Luc感染的培养物(示出7个克隆中的1个克隆的序列)的RNA,Ad11cDNA序列衍生自分离自wtAd11感染的培养物的RNA。序列编号是任意的。就Ad35wt序列而言,示出了wtAd35序列的第3339至3628位核苷酸。内含子序列(在cDNA序列中见到的一缺口)的两侧有剪接供体(SD)和剪接受体(SA)位点,与已知共有序列密切匹配。SeqMan软件将来自SD的前两个核苷酸(AG)放置在cDNA序列中的内含子序列的3’端而不是5’端。
图31:Ad35病毒中pIX加帽位点的位置。
Ad35中E1B基因和pIX序列附近的基因组组构的示意图;描述了pIX mRNA中转录起始位点和内含子边界。核苷酸序列为wtAd35
DNA(WO00/70071)。M=MunI,B=Bsu36I,SD=剪接供体,SA=剪接受体。转录起始位点(第3339位核苷酸,加帽位点),55K的终止密码子(TAA)及pIX的起始密码子(第3484位核苷酸,ATG)以黑体字标示。虚线标示未示出的序列。
图32:来自保留166bp的3’E1B序列的Ad35病毒的转基因PCR结果。
对Ad35.AdAptBsuLuc5Orf6(泳道1-9)和Ad35.AdAptBsuLuc病毒(泳道10-14)进行转基因PCR分析的结果示意图。M=1kb+标记(Invitrogen),P=pAdAptBsuLuc对照质粒。
发明概述
本发明揭示了一种重组B群腺病毒,其在接近E1B 55K的终止密码子的E1区中有一个缺失,与相似的Ad5重组腺病毒相比可以容纳较少的外源性序列。看起来当pIX编码区在E1B 55K终止密码子和pIX起始密码子之间的序列之后时,这是由于在给定的包装细胞中所述B群病毒中pIX基因的相对低表达所致。这示出当pIX启动子通过在这种病毒中包含E1B 55K编码区的序列或者通过使用一异源启动子以调节pIX而至少部分恢复时,可以使这种病毒更稳定和/或能容纳更多的外源性序列,由此在给定的包装细胞中达到正常表达或者甚至相对过表达。
本发明提供了一种重组腺病毒,其在E1区内具有至少一个缺失,所述重组腺病毒的特征在于在所述腺病毒中存在编码提高pIX基因表达的E1B 55K基因产物的序列的至少一部分,附带条件是所述重组腺病毒不表达功能性E1B 55K基因产物。这种腺病毒与缺少全部E1B编码序列的相应腺病毒相比更稳定和/或能携带更多的外源DNA。优选地,所述腺病毒包含位于pIX编码序列直接上游的大约700个或更少碱基对的序列。在优选的实施方案中,所述腺病毒是B群腺病毒,更优选的是衍生自或基于Ad35或Ad11的腺病毒。本发明进一步提供了提高在E1区中有至少一个缺失的重组腺病毒的稳定性和/或包装能力的方法,所述方法包括保留或再导入编码E1B 55K基因产物并提高pIX基因在所述腺病毒中表达的序列的至少一部分。除了存在提高pIX基因表达的E1B 55K序列之外,将pIX编码序列之前的序列改变为一更强的启动子也是可能的,从而提高pIX的表达,使得重组腺病毒稳定性提高和/或通过本发明方法生产的腺病毒颗粒的包装能力提高。因此本发明进一步提供了提高缺少至少E1区及包含外源遗传信息的重组腺病毒载体的稳定性和/或包装能力的方法和手段,所述方法包括当一种包装细胞中存在升高水平的pIX基因产物时在所述包装细胞中表达生产并装配所述重组腺病毒载体成病毒颗粒所需的元件的步骤,其中所述pIX基因产物的水平升高通过使用在所述载体中的一个修饰的pIX基因而过表达编码pIX蛋白的遗传信息而达到,所述修饰使pIX基因产物过表达。在一个优选的实施方案中,所述修饰的pIX基因包含一个异源启动子,其驱动编码pIX的遗传信息的表达,所述异源启动子是使所述编码pIX的遗传信息在所述包装细胞中过表达的一个启动子。在一个实施方案中,所述异源启动子至少部分衍生自或基于一种腺病毒血清型的pIX启动子,所述腺病毒血清型与所述重组腺病毒载体的内源近端pIX上游序列相比授予较高水平的pIX表达。在一个实施方案中,所述异源启动子至少部分衍生自或基于Ad5的pIX启动子。在一个方面,本发明提供了缺少至少E1区并包含感兴趣的基因的一种重组腺病毒载体,其中pIX基因被修饰,而且其中所述重组腺病毒载体不是衍生自腺病毒血清型5。本发明再一方面提供了包含一个修饰的腺病毒pIX基因的一种重组核酸序列,其中编码pIX蛋白的遗传信息不是衍生自腺病毒血清型5或腺病毒血清型7pIX编码序列。在一个优选的实施方案中,所述修饰的pIX基因包含驱动编码pIX的遗传信息表达的一个异源启动子。
本发明进一步提供了包含第一种血清型的腺病毒的结构和非结构元件的重组腺病毒载体,其中所述载体进一步包含编码E4-orf6蛋白的一个序列,其中所述序列选自以下一组中:a)衍生自与所述第一种血清型不同的第二种血清型的腺病毒的E4-orf6编码序列;b)通过一或多个密码子的缺失、突变、添加和/或取代衍生自所述第一种血清型的腺病毒的E4-orf6编码序列;和c)包含一种融合体的E4-orf6编码序列,所述融合体是衍生自与所述第一种血清型不同的第二种血清型的E4-orf6编码序列的一部分与衍生自第三种血清型的E4-orf6编码序列的一部分的融合体,其中所述第三种血清型与所述第一种血清型可以相同或不同。
本发明进一步提供了生产包含第一种血清型腺病毒的结构和非结构元件的这种重组腺病毒载体的方法,所述方法包括如下步骤:a)将腺病毒的必需元件提供给包含可表达形式的衍生自第二种血清型腺病毒的E1B-55K编码序列的互补细胞,以使得可以通过所述互补细胞装配所述重组腺病毒载体,其中所述元件包含与所述第二种血清型不同的所述第一种血清型腺病毒的至少一些结构和非结构元件及编码与所述互补细胞中所述可表达的E1B-55K蛋白相容的功能性E4-orf6蛋白或其功能部分、衍生物和/或类似物的一个序列;b)在生产和装配腺病毒载体的条件下在培养基中培养所述互补细胞;及c)从所述培养基和/或互补细胞中收获如此生产的重组腺病毒载体,其中编码相容的E4-orf6蛋白的序列存在于如此生产的重组腺病毒载体中。
本发明进一步提供了包含衍生自一种腺病毒的重组核酸分子的一种重组腺病毒,所述重组核酸分子在E1区有至少一个缺失,特征在于在所述重组核酸分子中存在编码E1B-55K基因产物的序列的至少一部分和/或pIX编码序列在一个异源启动子的控制之下,所述重组腺病毒进一步包含第一种血清型腺病毒的结构和非结构元件,其中所述重组腺病毒进一步包含编码功能性E4-orf6蛋白或其功能部分、衍生物和/或类似物的序列,其中所述序列选自以下一组:a)衍生自与所述第一种血清型不同的第二种血清型腺病毒的E4-orf6编码序列;b)衍生自包含一或多个密码子缺失、突变、添加和/取代的所述第一种血清型腺病毒的E4-orf6编码序列;及c)包含一个融合体的E4-orf6编码序列,所述融合体是衍生自第二种血清型的E4-orf6编码序列的一部分与衍生自第三种血清型的E4-orf6编码序列的一部分的融合体,其中所述第三种血清型与所述第一种血清型可以相同或不同。
本发明还提供了一种包装系统,其包含本发明的一种重组腺病毒及一种包装细胞,其中所述包装细胞及所述重组腺病毒共同包含在所述包装细胞中生产和装配所述重组腺病毒的所有必需元件,其中所述包装细胞表达编码与所述重组腺病毒的E4-orf6蛋白或其功能部分、衍生物和/或类似物相容的至少一种腺病毒E1B-55K蛋白或其功能部分、衍生物和/或类似物的核酸。
本发明还提供了生产一种包含第一种血清型腺病毒的结构和非结构元件的稳定重组腺病毒的方法,其中所述重组腺病毒包含衍生自一种腺病毒的重组核酸分子,所述核酸分子在E1区中有一个缺失,并且所述重组腺病毒包含衍生自编码E1B-55K基因产物的序列的至少一部分的核酸,所述基因产物提高pIX蛋白的表达,但不引起来自所述核酸分子的功能性E1B-55K蛋白的表达和/或具有在一个异源启动子控制下的pIX编码序列,所述方法包括如下步骤:a)将腺病毒的必需元件提供给表达可表达形式的衍生自第二种血清型腺病毒的E1B-55K编码序列或其功能部分、衍生物和/或类似物的互补细胞,以使得可以通过所述互补细胞装配所述重组腺病毒载体,其中所述元件包含与所述第二种血清型不同的所述第一种血清型腺病毒的至少一些结构和非结构元件及一种编码与所述互补细胞中所述可表达的E1B-55K蛋白相容的功能性E4-orf6蛋白或其功能部分、衍生物和/或类似物的序列;及b)在可以生产和装配重组腺病毒的条件下在培养基中培养所述互补细胞;及c)从所述培养基和/或互补细胞中收获如此生产的重组腺病毒。
本发明进一步提供了生产含有外源遗传信息的稳定腺病毒载体而不产生可复制的腺病毒的方法,其中所述载体是除了腺病毒5之外的血清型,所述方法包括如下步骤:在适于腺病毒载体生产的条件下培养细胞,所述细胞或其祖细胞已经被提供并表达编码腺病毒载体生产和包装所必需的所有元件的核酸,生产可复制的腺病毒载体所必需的元件存在于至少两个独立的核酸分子上而不导致分子间重组,所述必需元件包含来自除了Ad5之外的第一种血清型的腺病毒的至少一种决定血清型的结构元件和非结构元件,并包含来自与第一种血清型不同的第二种血清型的E1B 55K基因产物和与所述E1B 55K基因产物相容的E4 orf6基因产物。在某些实施方案中,编码腺病毒载体生产和包装所必需的所有必需元件的核酸包含一个重组pIX基因。在一个实施方案中,重组pIX基因是在一个异源启动子控制下的pIX编码序列。
另一方面,本发明提供了治疗或预防人体或动物疾病或失调的方法,所述方法包括为人或动物施用本发明的重组腺病毒载体。在其它方面,本发明提供了包含本发明腺病毒载体的疫苗和药物组合物。另一方面,本发明提供了本发明的重组腺病毒载体在制备预防或治疗人或动物疾病或失调的药物中的应用。本发明还涉及一种试剂盒,其包含本发明提供的细胞系和腺病毒载体以实践本发明提供的方法。
发明详述
本发明的一方面提供了提高在E1区有至少一个缺失的重组腺病毒的稳定性和/或包装能力的方法,所述方法包括以下步骤:相对于当pIX编码序列位于其没有E1B 55K序列的内源近端上游序列之后时含有的pIX基因产物的水平,当包装细胞中存在升高水平的pIX基因产物时,在所述包装细胞中表达生产及装配所述重组腺病毒成病毒颗粒所需的元件。在本发明方法的一些实施方案中,所述pIX基因产物的升高水平是通过在所述腺病毒中保留或再导入部分E1B 55K序列而带来的。在其它一些实施方案中,所述pIX基因产物的升高水平是通过在一种异源启动子控制下表达的pIX编码序列而带来的。本发明进一步提供了一种重组腺病毒,其包含在一种表达序列控制下的功能性pIX编码序列,所述表达序列包含能在给定包装细胞中增加pIX编码序列表达的部分E1B 55K序列,所述表达增加是相对于位于其没有所述部分E1B 55K序列的内源近端pIX上游序列之后的所述pIX编码序列的表达而言,条件是所述部分E1B 55K序列不编码功能性E1B 55K基因产物。本发明示出pIX启动子序列可以存在于所述E1B55K序列中,以及在所述表达序列中包括这些序列可以因此增加pIX表达。相对于所述pIX编码序列在其没有所述部分E1B 55K序列的内源近端上游序列之后时,所述E1B 55K序列的存在增加了所述重组腺病毒的稳定性和/或包装能力。
WO02/40665中描述了E1区具有缺失但具有完整E1B 55K编码区的血清型35腺病毒(pBr.Ad35.leftITR.ΔE1AΔ21K)。然而,一种功能性55K基因产物如存在于所揭示的载体中的产物抑制凋亡,因此希望的是获得缺少功能性EIB 55K表达的重组腺病毒,例如通过突变E1B 55K基因或优选地通过包括仅仅部分E1B 55K序列,更优选包括仅仅E1B 55K起始密码子下游序列。本领域技术人员清楚使所述载体中的E1B 55K序列的量最小化对产生具有最大E1缺失的病毒以容纳更多的外源核酸是有益的,同时保留足够的E1B 55K序列以具有提高重组腺病毒的稳定性和/或提高包装能力的益处。根据本发明的教导,本领域技术人员能发现E1B 55K内使重组腺病毒稳定和/或提高包装能力的最低限度需要的序列,例如通过使用标准分子克隆技术以获得从揭示的载体(pBr.Ad35.leftITR.ΔE1AΔ21K)开始的E1B 55K区域内的连续缺失,并确定稳定性和包装能力而进行。因此本发明的一个目的是提供一种重组腺病毒载体,其中所述编码E1B55K基因产物的序列包含正好位于pIX可读框直接上游的大约0.7kb或更少的腺病毒序列。在其它实施方案中,所述序列包含正好位于pIX可读框直接上游的腺病毒序列的不超过大约680、600、550、500、450、400、350、300、250、200、150或100个核苷酸。在一个实施方案中,这种重组腺病毒载体保留166bp的55K编码序列的3’端。本领域技术人员将清楚本发明非限于存在在天然腺病毒中pIX编码序列的直接上游邻近序列中发现的一个序列。根据本发明其也可以具有更上游的即来自E1B 55K区域的序列(例如一个限制或PCR片段,见例如实施例10和图9)与更近端的pIX调节序列融合,从而产生调节序列的一个人工组合,只要当与不存在任何E1B 55K序列时相对比时导致pIX在给定的包装细胞中表达提高即可。在一个优选的实施方案中,所述腺病毒是B亚群腺病毒,更优选是Ad35或Ad11腺病毒。血清型35或11的腺病毒已经示出特别适于施用给人体,因为与目前使用最多的血清型5相比其有少得多的个体具有这些血清型的中和抗体(WO00/70071)。本发明的另一方面提供了可作为本发明的腺病毒的基因组的核酸。
另外或者除了存在提高pIX基因表达的E1B 55K序列之外,也可以通过突变pIX基因,优选其启动子而过表达pIX自身,以提高重组腺病毒的稳定性和/或包装能力。一种修饰的pIX基因是具有不同启动子和/或转录终止子,和/或突变的编码序列例如通过密码子优化、导入稳定RNA的内含子等获得的突变序列的pIX基因。本发明的pIX基因包含编码pIX的遗传信息,并包括核酸如在天然腺病毒中发现的pIX基因、等位基因变体形式的编码突变pIX的cDNA及信息或者编码突变pIX的核酸,所述突变pIX具有至少部分pIX功能,其与正常pIX可以在定量或定性程度上不同,其衍生自或基于通过氨基酸突变、缺失、添加或移位或其组合而产生的pIX。
如果一种重组腺病毒缺失至少E1B 55K区域直至包括E1B 55K基因产物的终止密码子,pIX可读框将位于E1B 55K终止密码子和pIX的起始密码子之间的序列之后。这些序列在此称为“内源近端pIX上游序列”(例如基于Ad35的重组腺病毒载体中的Ad35pIX上游序列、基于Ad11的重组腺病毒载体中的Ad11pIX上游序列等在此方面称为内源性的;所述定义包括在天然发现的及不是在实验室中产生的等位基因变体;针对一些近端pIX上游序列见图3A所示)。
在此所用术语“异源启动子”是指与天然发现的pIX基因上游的序列,包括E1B 55K区域的序列及内源近端pIX上游序列不同的任何序列,并能作为启动子从而调节pIX编码序列的转录。一方面,异源启动子可以是至少部分衍生自或基于来自与重组腺病毒载体衍生自其中的血清型不同的另一种血清型腺病毒的近端pIX上游序列(即E1B 55K终止密码子与pIX的起始密码子之间的序列)的一个序列(例如Ad35重组腺病毒载体中Ad5pIX上游序列)。在此将其称为“非内源性近端pIX启动子”。
在本发明的一个实施方案中,Ad35衍生的pIX表达由Ad5非内源性近端pIX启动子驱动。可以使用衍生自或基于来自与腺病毒载体的内源性pIX近端序列相比授予较高水平pIX表达的血清型的近端pIX上游序列的任何非内源性近端pIX启动子。这种非内源性近端pIX启动子的鉴别可以基于序列信息,如本领域技术人员从实施例4中显而易见的信息。在特殊的实施方案中,本发明的腺病毒载体衍生自或基于腺病毒亚群E血清型,或者优选亚群B血清型。在特异的实施方案中,所述腺病毒载体衍生自或基于腺病毒血清型35(Ad35)、Ad11、Ad7或Ad4。在另外的实施方案中,所述非内源性近端pIX启动子是至少部分衍生自或基于亚群C、A、D或F的腺病毒的近端pIX上游序列。在特殊的实施方案中,所述非内源性近端pIX启动子至少部分衍生自或基于腺病毒血清型12(Ad12)、Ad9或Ad40,或者更优选Ad5或Ad2的近端pIX上游序列。或者,作为非内源近端pIX启动子的序列可以通过本领域技术人员已知的普通分子学方法根据经验发现,如通过转录分析进行,其中可以常规测试启动子强度。本领域技术人员清楚来自启动子的元件可以交换而不用交换整个启动子,例如添加、缺失或突变已知的启动子的转录因子结合序列可以影响其强度。突变至少一部分启动子序列可以通过突变对序列加以改变而进行,如通过添加、缺失或交换一或多个核苷酸,包括具有已知功能的核苷酸序列而进行。取代启动子序列通过一个不同的启动子置换部分或全部这些序列而进行。这种置换可以根据本领域技术人员熟知的标准分子生物学技术进行。可以构建与选择的pIX基因可操纵相关的任何启动子并测试其作用。
另一方面,一种异源启动子与腺病毒非内源近端pIX启动子不相关。因此,异源启动子也可以是病毒启动子,包括但非限于基于或衍生自巨细胞病毒(CMV,例如人 CMV立即早期基因启动子,在此称作CMV启动子)、劳斯肉瘤病毒(RSV,例如RSV长末端重复启动子,在此称作RSV启动子)、TK、HBV、SV40等的启动子。在一个实施方案中,一种腺病毒E1B启动子用作异源启动子。细胞启动子也可以用作异源启动子,这些启动子包括但非限于来自PGK、金属硫蛋白、EF1-α、β-肌动蛋白等的启动子。本发明也可以使用包含来自一个以上启动子的元件的合成的或杂合启动子,这些启动子均包含在称为异源启动子的范围内。所用的启动子可以是组成型或可诱导启动子。就可诱导启动子而言,如果一种启动子在其被诱导状态提供过表达则认为其适于本发明。导致本发明的pIX过表达的任何启动子序列均可用作本发明的异源启动子。除了启动子强度之外,另一方面确定一特殊启动子当存在于重组腺病毒载体中时的效力是其大小,较长的启动子将占据转基因可利用的空间。本领域技术人员能使用本发明针对启动子长度和强度考虑到插入体大小而用实验方法优化载体,以发现最稳定的重组载体。
显而易见异源启动子仍可以含有或包括部分或全部内源近端pIX上游序列,或者全部置换这些序列,只要根据本发明的异源启动子可以引起编码pIX的遗传信息在选择的包装细胞中过表达即可。本领域技术人员也清楚当一内源非pIX腺病毒启动子(例如E1A、E2A、等启动子)或例如调节转基因的一异源启动子通过使用非pIX腺病毒基因或转基因与pIX编码序列(以任一顺序)之间的内部核糖体进入位点(IRES)用于驱动pIX表达时,根据本发明认为该启动子包含在称为“异源启动子”的范围内。
本发明中pIX基因产物水平的升高或增加是pIX基因在选择的包装细胞中过表达的结果。在此所述pIX基因的过表达是指在给定的包装细胞中pIX的表达水平在RNA或蛋白质水平或这两个水平上均高于当pIX的编码区位于内源近端pIX上游序列之后时获得的pIX表达水平。在本发明中“过表达”是指一腺病毒的特定异源启动子-pIX组合与选择的特定包装细胞的组合的过表达。确定表达水平的方法是本领域技术人员熟知的,包括但非限于RT-PCR、Northern印迹、Western印迹等。就本发明而言,测定过表达通过确定在选择的给定包装细胞中具有给定的插入体(例如荧光素酶)的重组腺病毒中的表达水平,其中编码pIX的遗传信息在没有E1B 55K序列的内源近端pIX上游序列之后,并将这些表达水平与在重组腺病毒中的那些表达水平相对比,其除了pIX编码区由异源启动子或来自内源E1B 55K基因的序列(其天然启动子已经至少部分被重构)调节外其它都一样。pIX的过表达是以高于1的比率表示的,所述比率是通过异源或天然启动子获得的表达水平与通过无E1B 55K序列的内源近端pIX上游序列获得的表达水平的比率。包装细胞的选择通过如与包装细胞中与互补腺病毒功能相互作用的重组腺病毒的元件的血清型、产物纯度(如没有来自产生的批次物中的能复制的腺病毒)、使用便利性、生长特性等因素加以确定。本领域技术人员已知包装细胞的实例,包括在此使用的293细胞、911细胞和PER.C6TM细胞以及其适于互补特异血清型的腺病毒载体的衍生物如PER55K。
应清楚在本发明的所有实施方案中,pIX编码序列及驱动pIX表达的调节序列可以位于腺病毒基因组中其天然位置上,以及在腺病毒基因组的不同部分中,例如在最初含有E3序列的区域中。
稳定性提高的重组腺病毒能将较大的基因组掺入病毒颗粒中(病毒体)。因此,提高在此使用的重组腺病毒的稳定件使得本发明的重组腺病毒可以包括更多的外源遗传信息,包括感兴趣的基因。另外,根据本发明的稳定提高的重组腺病毒能增殖更多代而无不稳定的迹象。稳定性可以通过本领域技术人员已知的一些方法测定,包括但非限于对重组病毒进行PCR以证明希望的重组腺病毒载体的存在。不稳定将产生副产物,其也可以通过PCR等方法观测到。也可以使用病毒DNA的限制分析、确定病毒颗粒的相对感染性、确定腺病毒颗粒的热稳定性等确定重组腺病毒载体的稳定性(Caravokyri andLeppard,1995)。确定重组腺病毒载体的稳定性/不稳定性的方法见本申请的实施例3所述。在此使用的一种重组腺病毒,也称作重组腺病毒载体或腺病毒载体,衍生自或基于一种腺病毒,缺少腺病毒的至少部分E1区(包含E1A和E1B基因)并可以包含希望通过所述载体输送和/或表达的外源遗传信息。在此所用的外源遗传信息是非天然存在于腺病毒中的任何遗传信息,也称作转基因。这包括但非限于感兴趣的基因、表达盒等。这种外源遗传信息可以充填基因组中已经通过腺病毒E1序列的缺失而可利用的空间。重组腺病毒载体用于多种目的,如在基因治疗、疫苗制备等应用中。除了E1区缺失之外,E3序列也从这种腺病毒载体中缺失,以在某些优选的实施方案中提高对外源遗传信息的容量。当需要复制腺病毒载体时,如果需要与包含腺病毒载体中缺少的遗传信息的包装细胞组合,可以使用组合E1缺失的其它缺失及E2、E3和E4区的部分或完全缺失的多种组合。具有组合了腺病毒基因组中任选任何其它缺失的E1区缺失的所有重组腺病毒包括在本发明范围内。本发明的腺病毒可以用于不同方案中,如基因治疗或预防和/或治疗性免疫接种,包括肿瘤免疫接种和抗病毒免疫接种。为此,腺病毒载体作为基因输送载体,其中将一种非天然基因掺入腺病毒基因组中。该腺病毒颗粒随后可特异性定向于感兴趣的靶细胞;腺病毒通过壳体-受体结合或者通过其它方式与特异细胞结合,并输送转基因。腺病毒的定向可以以许多不同方式进行。腺病毒载体定向领域的技术人员知道用于将腺病毒载体输送于感兴趣的细胞的所有不同的可能性。这种可能性包括但非限于壳体改变(尾丝、六邻体和/或五邻体修饰,如缺失,不同血清型的尾丝之间的交换,及添加肽和/或其它结合组分),其中产生了识别感兴趣的细胞上存在的受体的嵌合尾丝,或者其中利用了基于五邻体的结合。其它可能性是将定向组分与壳体蛋白连接,其中例如将结合肽、已知的强结合蛋白或者抗体或其一部分与壳体蛋白连接以实现特异性定向。这种载体均可使用本发明提供的方法和手段生产。
因此,本发明还揭示了根据本发明的重组腺病毒载体,其进一步包含编码非腺病毒蛋白的一个序列。这种序列可以存在于腺病毒主链内的不同位置上,但优选位于本发明的重组腺病毒载体中缺少的E1区中。E1区由互补细胞中存在的互补元件互补。启动子、转基因及其它调节序列的方向可以朝向左侧以及右侧反向末端重复。
本发明还用于生产基于腺病毒和/或其它病毒如腺伴随病毒(AAV)的病毒载体,其中如Ad-AAV嵌合病毒的组合可以整合进宿主细胞基因组中。本领域已知产生整合的腺病毒的一些方法。通常地,本发明也用于生产可(特异性或非特异性)整合的腺病毒形式。
正如所提及的,一些非腺病毒转基因可以克隆进本发明的重组腺病毒载体中。这些不仅包括调节性核酸序列如增强子、启动子(例如强非腺病毒启动子如巨细胞病毒启动子、SV40启动子及RSV启动子)及聚腺苷酸化信号,也包括针对治疗目的的异源基因。因此,在本发明的一个方面中,提供了本发明的重组腺病毒载体,其中所述非腺病毒蛋白选自以下一组:细胞死亡诱导多肽、肿瘤特异性抗原、病毒蛋白、激素和细胞因子。非腺病毒因子、蛋白质、多肽和肽的非限制性实例是转录因子、细胞内信号化蛋白、磷酸酶、激酶、凋亡抑制因子、受体拮抗剂、可溶形式的膜结合受体、RNA抑制剂、反义RNA′s、诱饵因子(decoy factors)、核酶,及更特异性地,胸苷激酶、促红细胞生成素、新红细胞生成刺激蛋白(NESP)、IL3、ceNOS、γ-干扰素和gp100。就免疫接种目的而言,非腺病毒病毒蛋白可以克隆进通过本发明的方法和手段提供的重组腺病毒载体中。这种病毒蛋白包括但非限于针对HIV疫苗的gag、pol、env、nef等,针对人乳头状瘤病毒疫苗的E6和E7蛋白,针对疟疾疫苗的来自疟原虫(Plasmodiumprotozoa)的环子孢子蛋白,针对轮状病毒疫苗的轮状病毒成分,针对埃博拉病毒(ebola)疫苗的埃博拉病毒蛋白,针对呼吸道合胞病毒疫苗的来自呼吸道合胞病毒的F和G基因产物,针对流感疫苗的HA和NA等。
根据本发明的腺病毒优选是人腺病毒,即衍生自或基于能感染人细胞的腺病毒,但本发明同样用于非人腺病毒。本领域技术人员知道除了所有人腺病毒之外,本领域已经鉴别了许多非人腺病毒。明显地,非人腺病毒也可以用于达到本发明揭示的相同结果。使用本发明的方法和手段可以生产的非人腺病毒的非限制性实例是狗、牛、猴和鸟腺病毒。在此所用的血清型因此超出物种限制性血清型。
在此所用术语“衍生自”是指在腺病毒中正常发现的核酸序列、基因或蛋白质用于产生根据本发明的重组腺病毒。本领域技术人员熟知产生这种重组腺病毒的方法,包括但非限于普通分子生物学方法如利用限制酶将遗传信息克隆进希望的constellations中的方法等。重组腺病毒也可以基于腺病毒序列。“基于”是指包括基于对这种遗传信息的了解合成构建遗传信息。这种方法包括但非限于使用腺病毒遗传物质作为PCR模板以构建一种基于所述模板腺病毒的序列的新的腺病毒构建体,构建具有希望序列的完全合成的遗传信息,例如通过将合成的寡核苷酸与希望的构建体连接而进行等。应理解“衍生自”非必需意味着野生型DNA的直接克隆。本领域技术人员也知道获得某一核酸的突变形式的分子生物学可能性。这些突变可以导致不同的功能性,但它们在一种方式中也可以是沉默的,即在某些突变不改变特定DNA及其编码的蛋白质的功能性的方式中。因此,术语“其功能部分、衍生物和/类似物”应理解为与其相关的核酸的等同物。本领域技术人员意识到某些缺失、交换、(点)突变、添加等也可以产生与原始核酸具有相似功能的核酸。因此应理解不显著改变蛋白质如pIX蛋白、E4-orf6和E1B-55K基因产物的功能这种变化在本发明的范围内。本领域技术人员清楚获得编码重组腺病毒的遗传信息的方法在不偏离本发明的范围内可以变化。
人腺病毒分为A-F亚群,其涵盖了51个血清型(见例如EP0978566所述)。就一些应用而言,使用衍生自或基于来自特异亚群或来自针对希望的细胞类型例如树突细胞具有组织向性的某些血清型的腺病毒的腺病毒载体可以是有益的(WO02/24730)。在种群中一般没有抗来自某些亚群或来自一特异性血清型的腺病毒的中和抗体对决定选择的血清型也是一个重要参数(WO00/70071)。由于亚群B病毒之间的相似性(见例如实施例4和11),期望本发明特别适用于亚群B的腺病毒。因此,本发明优选的实施方案涉及衍生自或基于亚群B中分类的腺病毒的腺病毒载体。人腺病毒的亚群B包含Ad3、Ad7、Ad11、Ad14、Ad16、Ad21、Ad34、Ad35和Ad50。本发明优选的实施方案涉及衍生自或基于Ad35或Ad11血清型的重组腺病毒载体。除了针对特异性应用而选择血清型之外,可以使用所谓的嵌合腺病毒。这些嵌合腺病毒包含来自一或多个腺病毒血清型的编码包被蛋白如尾丝、五邻体或六邻体的部分或全部遗传序列,与来自其它血清型的剩余遗传信息(主要腺病毒载体部分)连接,其可用于降低主要腺病毒载体的免疫原性或改变主要腺病毒载体的组织向性(EP0978566)。在此所用术语“主要”是指其为所述嵌合病毒提供大多数遗传信息,嵌合腺病毒因此包含在这种病毒的主要部分的血清型组中。本领域技术人员清楚当这些嵌合病毒面临相似的不稳定性问题时,本发明也可以用于这种嵌合腺病毒。可以例如期望包含Ad35序列作为主要部分并包含衍生自或基于例如Ad11腺病毒的尾丝的嵌合腺病毒在增殖方面与针对Ad35重组腺病毒载体所报道的具有相似的不稳定性。因此,当在本申请中提及重组腺病毒时,嵌合腺病毒载体包含在本发明范围内。
本领域技术人员熟知生产和装配重组腺病毒载体所必需的元件(如前述;USpatent 5,994,128;Russell,2000)。以病毒体形式生产E1缺失的重组腺病毒是在包装细胞中进行的,该细胞也称作互补细胞。这种细胞反式(in trans)提供在所述重组腺病毒中缺少的生产重组病毒(重组病毒体)所必需的腺病毒的遗传信息。熟知的包装细胞是293细胞、911细胞和PER.C6TM细胞(如前)。就大多数目的而言,优选使用一种包装细胞和缺少引起同源重组而产生能复制的腺病毒的重叠序列的一种重组腺病毒载体(US patent 5,994,128)。在Centerfor Applied Microbiology and Research的European Collection ofAnimal Cell Cultures以保藏号96022940保藏的PER.C6TM因此是增殖重组腺病毒的一种非常合适的包装细胞。也已经设想减少产生能复制的腺病毒的其它方法,涉及例如操纵腺病毒序列以降低包装细胞和载体中存在的序列之间的同源性(例如Hehir et al.,1996;Robert etal.,2001所述)。除了必需的E1区之外,包装细胞还可包含其它腺病毒序列以在当在所用的重组腺病毒中功能性缺少例如部分或全部E2、E4等时互补其它腺病毒功能。包装细胞中的互补信息可以整合在基因组中,或者作为染色体外拷贝存在于例如质粒、载体、粘粒等上。其它方法利用所谓的辅助病毒,其包含在重组腺病毒中缺少的遗传信息。重组腺病毒载体也用作所谓的辅助病毒,用于生产含有缺失大部分或全部腺病毒基因的基因组的重组腺病毒(gutless载体或辅助依赖性腺病毒)。在这种gutless腺病毒的最后生产中,需要避免辅助腺病毒的包装。本领域技术人员熟知实现这种目的的方法,例如在包装信号中遗传工程化的位点上使用位点特异性重组酶以缺失这种包装信号。通常必需使用CsCl梯度分离从希望的gutless病毒中分离剩余的辅助病毒。当辅助和gutless病毒的基因组长度最大程度不同时,易于达到这个目的。因此,优选使用大的而不是小的辅助病毒。本领域技术人员清楚本发明可同样用于提高重组腺病毒的稳定性,通过使用具有升高的pIX表达的重组辅助病毒进行,这可以通过本发明中所述的方法实现。含有可用于互补重组腺病毒的遗传信息以产生重组病毒颗粒的任何细胞,包含在包装细胞的范围内。本领域技术人员清楚通过本发明获得的优势非依赖于所用的包装细胞。
编码pIX的遗传信息可以存在于重组腺病毒载体上,也可以不依赖于所述重组腺病毒载体而存在,这种病毒外遗传信息(extraviralgenetic information)可以整合进基因组中或者作为染色体外拷贝存在于例如质粒、载体、粘粒等之上。将遗传信息导入包装细胞中可以根据多种方法进行,如通过lipofectamin转染、磷酸钙沉淀、病毒感染等方法进行。本领域技术人员熟知这种方法,而且用于导入遗传信息的方法就本发明范围不是关键的。本文所用的可表达形式的功能性pIX是指编码pIX的遗传信息,其与能驱动所述编码pIX的遗传信息在包装细胞中表达的启动子或其它调节序列可操纵地连接。将遗传信息导入包装细胞可以在导入重组腺病毒载体之前、同时或之后进行。发现pIX在基于293的包装细胞系中的组成型附加型表达互补pIX突变体腺病毒类型5的缺乏(Caravokyri and Leppard,1995)。然而,就这种应用而言,需要含有EBNA1表达盒的特殊附加型质粒,而且这种细胞系中腺病毒载体的增殖经受部分附加体非常可能成为重组腺病毒载体一部分的不利状况。因此,在本发明优选的实施方案中,编码功能性pIX的遗传信息存在于腺病毒载体上。
在减少产生能复制的腺病毒的重组量的尝试中,一些作者使用衍生自由Ad5的突变的pIX启动子驱动的B群病毒Ad7的pIX基因以消除(含有Ad5衍生的序列)包装细胞与重组腺病毒载体的核酸之间的重叠(Robert et al.,2001)。然而,那些实验未提高病毒载体的稳定性,而且在那些实验中未测定这种稳定性。在本发明中,腺病毒载体中pIX的转录调节序列根据提高病毒的稳定性和/或提高病毒体中外源遗传物质的容量的目的而加以改变。本发明证明了衍生自Ad35的重组腺病毒载体与具有内源(即Ad35衍生的)近端pIX上游序列的相应病毒相比更稳定/可携带更多外源遗传信息,所述Ad35包含在衍生自Ad5的Ad5衍生近端pIX启动子控制下的pIX基因。因此,本发明还提供了包含功能性pIX编码序列并在E1区中有至少一个缺失的一种重组腺病毒,其中pIX编码序列在异源启动子的控制下,而且其中所述重组腺病毒衍生自或基于除了腺病毒血清型5之外的腺病毒。在某些实施方案中,所述异源启动子是一种非内源近端pIX启动子。在优选的实施方案中,编码pIX的遗传信息衍生自或基于Ad35或Ad11。在这种实施方案中,优选的非内源pIX启动子是Ad5启动子。
另一方面,本发明提供了可通过本发明的方法获得的一种重组腺病毒载体。这种重组腺病毒载体用于例如制备疫苗(WO00/70071;WO01/38362;WO02/24730),作为基因输送载体等。
针对这种重组腺病毒载体选择一种希望的主要血清型可以用于获得与常用的Ad5腺病毒载体相比组织向性改变的载体,和/或比这种Ad5衍生的载体免疫原性低而可以被使用(WO00/70071)。为产生重组腺病毒载体,将转基因克隆进含有缺少缺失E1序列的腺病毒左侧部分和具有进行克隆的限制酶位点的一个质粒(衔接质粒(adapterplasmid))中。然后通过包含具有5’端重叠序列的腺病毒右侧部分的粘粒与衔接质粒的3’端同源重组而产生重组腺病毒载体(见本申请的实施例;WO99/38362中所述方法)。因此本发明的另一方面提供了一种重组核酸序列,其包含腺病毒左侧ITR,包装信号、E1区缺失的其它腺病毒序列、至少部分E1B 55K可读框及pIX编码序列。本发明的再一方面提供了一种修饰的腺病毒pIX基因,其中编码pIX蛋白的遗传信息不是衍生自腺病毒血清型5或腺病毒血清型7pIX编码序列。在优选的实施方案中,所述修饰的pIX基因包含一个异源启动子。本发明还涉及一种药物组合物,其包含根据本发明的或通过本发明的方法获得的一种重组腺病毒载体。所述药物组合物进一步包含一种可接受的药物载体,通常由制备药物领域的技术人员所应用。另外,本发明涉及一种治疗人体的方法,包括为人体施用本发明的重组腺病毒载体或者施用由本发明提供的一种药物组合物。
本发明进一步提供了一种重组腺病毒包装细胞,其包含根据本发明的一种重组腺病毒。在一个实施方案中,所述重组腺病毒包装细胞包含能互补所述重组腺病毒的E1B 55K缺乏的核酸,其中所述重组腺病毒包含的核酸分子包含编码提高pIX基因表达的E1B 55K基因产物的序列的一部分,附带条件是后者重组核酸分子不编码功能性E1B 55K基因产物,其中所述细胞和所述重组腺病毒不包含导致包含编码功能性E1B 55K蛋白的核酸的重组腺病毒形成的序列重叠。这个实施方案特别适用于防止包含额外的腺病毒功能的重组腺病毒的形成。
本发明进一步揭示解决与在Ad包装/互补细胞中减少非C群腺病毒载体的互补相关的某些难题的方法和手段。尽管在Ad5互补细胞系中存在功能性Ad5E1B-55K表达,但发现当腺病毒主链是非C群腺病毒来源时仅可以产生非常低滴度的腺病毒载体;这个发现意味着E1B-55K与另一种(病毒)蛋白的相互作用中的血清型特异性。本发明揭示了这种血清型依赖性可以通过提供与互补细胞系提供的E1B-55K蛋白相容的E4-orf6蛋白而避免。正如本发明所揭示的,E1B-55K和E4-orf6形成参与抑制将细胞mRNA从核转运至胞质的一种复合物,该复合物也参与刺激病毒mRNA从核转运至胞质(参见Leppard 1997和1998所述)。本发明人已经观测到包装细胞中病毒载体的适当互补需要存在相容的E1B-55K和E4-orf6基因产物。如果包装细胞包含编码互补应包装的载体未提供的功能的蛋白质的某些序列,则包装细胞也称作互补细胞。在此所用术语“相容“是指可利用的E1B-55K基因产物之间的一种复合物能与可利用的E4-orf6基因产物形成一种功能性复合物,在某种意义上这种蛋白复合物支持病毒复制、增殖和/或包装为与野生型情况相似或者与当重组腺病毒血清型5载体在Ad5互补细胞系如293或PER.C6TM上生产时发现的情况相似的水平。如果在其中病毒形成的生产期间包装细胞包含相容的至少E1B-55K蛋白和E4-orf6蛋白,则在包装细胞中的载体复制是有效的。优选地,E1B-55K和E4-orf6序列来自相同腺病毒亚群(如A、B、C、D、E或F)内的腺病毒。更优选地,E1B-55K和E4-orf6序列来自相同血清型。由于本领域可利用能支持亚群C如血清型5的腺病毒生长的确立细胞系,甚至更优选E1B-55K和E4-orf6基因衍生自腺病毒血清型5。本领域技术人员知道相容性可以在腺病毒载体生产领域的技术人员已知的互补测试或分析中确定。本领域技术人员也知道本发明也可用于在任何互补细胞系上生产任何腺病毒血清型,只要E1B-55K和E4-orf6蛋白是相容的即可。
进一步观测到与互补细胞系中E1B“匹配”的E4-orf6基因产物可以由腺病毒载体提供,通过将选择的腺病毒载体中的E4-orf6用与包装细胞系中存在的E1B基因相容的E4-orf6编码序列置换而进行。令人惊奇地发现这种修饰对载体的稳定性、复制、包装、装配和生产无不利作用。
本发明的一特异方面是现在能在通常用于生产腺病毒血清型5或者亚群C的其它血清型如血清型1、2和6的细胞系上有效生产与亚群C的那些血清型不同的腺病毒血清型。本发明提供了生产非C群腺病毒而不需要单独提供具有E4-orf6的互补(包装)细胞的方法,因为与互补E1B-55K序列相容的E4-orf6序列掺入腺病毒主链中。
本发明提供了包含第一种血清型的腺病毒的结构和非结构元件的一种重组腺病毒载体,其中所述载体进一步包含编码功能性E4-orf6蛋白的一个序列或其功能部分、衍生物和/或类似物,其中所述序列选自以下一组:a)衍生自与所述第一种血清型不同的第二种血清型的腺病毒的E4-orf6编码序列;b)衍生自包含一或多个密码子的缺失、突变、添加和/或取代的所述第一种血清型的腺病毒的E4-orf6编码序列;及c)包含一个融合体的E4-orf6编码序列,所述融合体是衍生自与所述第一种血清型不同的第二种血清型的E4-orf6编码序列的一部分与衍生自第三种血清型的E4-orf6编码序列的一部分的融合体,其中所述第三种血清型与所述第一种血清型可以相同或不同。在一个实施方案中,本发明提供了一种重组腺病毒载体,其中所述第一种血清型与所述第二种血清型来自不同的腺病毒亚群。在一个优选的实施方案中,提供了一种本发明的重组腺病毒载体,其中所述第一种血清型来自除了亚群C之外的一个亚群,而且其中所述E4-orf6编码序列衍生自亚群C的腺病毒血清型。更优选本发明的重组腺病毒,其中所述第一种血清型来自亚群B,所述第二种血清型来自亚群C。甚至更优选地,所述E4-orf6编码序列衍生自腺病毒血清型5。本发明的重组腺病毒包含结构和非结构元件。结构元件的实例是编码壳体蛋白如尾丝、六邻体和五邻体蛋白的基因以及其自身基因产物。非结构元件的实例是早期基因,其在感染细胞时表达而且当感染周期进行时被下调。非结构元件的其它实例是编码在复制期间活性蛋白质的基因,如pol和pTP。
核酸中的一些变化如缺失、突变、添加和/或取代一或多个密码子可以显著改变编码的基因产物的结构和/或功能性。本发明因此还涉及E4-orf6编码序列,其衍生自与携带例如结构和非结构元件的基因的主链相同的腺病毒血清型,但其中E4-orf6编码序列已经突变由此其变得与生产腺病毒载体的互补细胞中存在的E1蛋白(如E1B-55K基因产物)相容。可以将密码子完全改变以改变编码的氨基酸,但其也可以部分突变以改变编码的氨基酸。核酸的缺失可以导致丧失一或多个编码的氨基酸,同时这也可以导致移码。本发明还涉及腺病毒核酸中存在的E4-orf6序列,其包含衍生自不同血清型的不同部分,其中可以使用来自一个血清型的使蛋白质在相容性中起作用的结构域,而E4-orf6序列的剩余部分或其一部分衍生自另一种(不)相关的血清型(例如来自相同亚群,来自不同亚群或者来自不同物种或其组合)。因此应用相容的E4-orf6融合蛋白也在本发明的范围内。这种融合蛋白可以是一些核酸的产物。
本领域技术人员意识到除了所有人腺病毒之外,本领域还鉴别许多非人腺病毒。显然也可以应用非人腺病毒以达到与本发明揭示的相同结果。技术人员清楚E1B-55K与E4-orf6之间的相容性可以非限于人腺病毒,而且来自特异于不同物种的腺病毒的元件也可以是相容的。因此,本发明的另一方面是可以在本领域可利用的已知包装细胞系上生产高滴度的非人腺病毒,只要E1B-55K与E4-orf6基因产物是相容的即可。使用本发明的方法和手段可以生产的非人腺病毒的非限制性实例是狗、牛、绵羊、青蛙、猪、马、猴和鸟类腺病毒。因此在此所用的血清型超出物种限制性血清型。如果例如一种猴腺病毒E4-orf6基因产物与包装细胞提供的E1B-55K相容,则这种组合包含在本发明范围内。同样,当不同的血清型或者衍生自例如与包装细胞的E1B基因相容的人和鸟类腺病毒的E4-orf6序列之间的融合体应用时,则这种特殊组合也包含在本发明范围内。
本发明提供了一种生产包含第一种血清型的腺病毒的结构和非结构元件的重组腺病毒载体的方法,所述方法包括如下步骤:a)将腺病毒的必需元件提供给携带可表达形式的衍生自第二种血清型腺病毒的E1B-55K编码序列或其功能部分、衍生物和/或类似物的互补细胞,以使得可以通过所述互补细胞装配所述重组腺病毒载体,其中所述元件包含与所述第二种血清型不同的所述第一种血清型的至少一些结构和非结构元件及编码功能性E4-orf6蛋白的一个序列或其功能部分、衍生物和/或类似物,其与所述互补细胞中所述可表达的E1B-55K蛋白相容;b)在生产和装配腺病毒载体的条件下在培养基中培养所述互补细胞;及c)从所述培养基和/或互补细胞中收获如此生产的重组腺病毒载体,其中编码相容的E4-orf6蛋白的序列存在于如此生产的重组腺病毒载体中。
在本发明的一个方面,提供了一种根据本发明的方法,其中所述E4-orf6编码序列选自以下一组:i)衍生自所述第二种血清型的腺病毒的E4-orf6编码序列;ii)衍生自与所述第一种和第二种血清型不同的第三种血清型的腺病毒的E4-orf6编码序列;iii)衍生自包含缺失、突变、添加和/或取代一或多个密码子的所述第一种血清型的腺病毒的E4-orf6编码序列;及iv)包含一个融合体的E4-orf6编码序列,所述融合体是衍生自第三种血清型的E4-orf6编码序列的一部分与衍生自所述第二种血清型腺病毒的E4-orf6编码序列的一部分的融合体,其中所述第三种血清型与所述第一种血清型可以相同或不同。在一个优选的实施方案中,所述第一种和所述第二种血清型来自不同的亚群。在一个更优选的实施方案中,所述第二种血清型是亚群C的腺病毒血清型。在一个更优选的实施方案中,所述第二种血清型是腺病毒血清型5。在本发明的其它特殊方面中,所述第一种血清型是亚群B的腺病毒血清型。优选地,所述第一种血清型选自由腺病毒血清型11、14、16、21、34、35和50组成的组中。
本领域已知用于互补重组腺病毒载体的一些包装细胞以生产、装配和包装腺病毒颗粒。这种细胞系的非限制性实例是HEK-293、911和PER.C6TM细胞。优选使用经证实输送高滴度腺病毒原种的细胞系。这种细胞系以稳定形式表达E1蛋白,这因此是本发明使用细胞系和方法的一个优选方面,其中所述E1B-55K编码序列整合进所述互补细胞的基因组中。更优选的是衍生自原代二倍体人体细胞或其祖细胞的互补细胞。甚至更优选地,所述互补细胞衍生自原代人成视网膜细胞、原代人胚胎肾细胞、原代人神经元细胞或者原代人羊膜细胞(primary human amniocyte)。特别优选的是互补细胞在本发明的方法中的应用,其中所述互补细胞是PER.C6TM细胞或其衍生物。本领域熟知当使用与PER.C6TM细胞提供的核酸不重叠的腺病毒DNA时,PER.C6TM细胞不产生能复制的腺病毒。本领域使用的许多腺病毒载体缺少E1区,因此在本发明的一个方面中,所述互补细胞包含整合进其基因组的编码至少一种腺病毒E1A蛋白的核酸。优选地,所述编码至少一种腺病毒E1A蛋白的核酸衍生自与亚群B不同的亚群的腺病毒血清型。更优选地,所述编码至少一种腺病毒E1A蛋白的核酸衍生自亚群C的腺病毒血清型。特别优选的实施方案是其中所述编码至少一种腺病毒E1A蛋白的核酸衍生自腺病毒血清型5。在本发明的另一个实施方案中,提供了一种方法,其中所述E4-orf6编码序列及所述E1B-55K编码序列衍生自不同的腺病毒血清型,其中所述不同的腺病毒血清型是相同腺病毒亚群的成员。优选地,所述E4-orf6编码序列和所述E1B-55K编码序列衍生自不同的腺病毒血清型,其中所述不同的腺病毒血清型均是亚群C的成员。更优选地,所述E4-orf6编码序列和所述E1B-55K编码序列衍生自相同的腺病毒血清型。特别优选这样的方法,其中所述E4-orf6编码序列和所述E1B-55K编码序列衍生自腺病毒血清型5。
本发明还涉及这样的方法,其中腺病毒载体可以使用适当的互补/包装细胞和感兴趣的腺病毒载体生产。为进行一种有效的生产程序,应用具有适当腺病毒载体的正确细胞是有益的。因此,本发明还涉及一种试剂盒(kit of parts)(也称为包装系统),其包含:a)生产包含第一种血清型腺病毒的结构和非结构元件的重组腺病毒载体的互补细胞,所述细胞携带可表达形式的衍生自第二种血清型腺病毒的E1B-55K编码序列或其功能部分、衍生物和/或类似物;及b)一或多个可复制的核酸载体,所有必需的腺病毒元件如可以通过所述互补细胞装配所述重组腺病毒载体,其中所述元件包含来自与所述第二种血清型不同的所述第一种血清型的腺病毒的至少一些结构和非结构元件及一个编码与所述互补细胞中所述可表达的E1B-55K蛋白相容的功能性E4-orf6蛋白或其功能部分、衍生物和/或类似物的序列。优选地,使用一种试剂盒,其中所述E4-orf6编码序列选自以下一组:a)衍生自所述第二种血清型的腺病毒的E4-orf6编码序列;b)衍生自与所述第一种和第二种血清型不同的第三种血清型的腺病毒的E4-orf6编码序列;c)衍生自包含一或多个密码子的缺失、突变、添加和/或取代的所述第一种血清型的腺病毒的E4-orf6编码序列;及d)包含一个融合体的E4-orf6编码序列,所述融合体是衍生自第三种血清型的E4-orf6编码序列的一部分与衍生自所述第二种血清型腺病毒的E4-orf6编码序列的一部分的融合体,其中所述第三种血清型与所述第一种血清型可以相同或不同。
本发明特别用于复制E1缺失的嵌合腺病毒,其几乎全部衍生自除了腺病毒5之外的血清型。这种载体仅需要为其提供编码腺病毒5E4orf6或其功能部分、衍生物和/或类似物的核酸。一旦如此,该载体可以在正常腺病毒5E1互补包装细胞系上有效复制。该载体的稳定性得以改良,而且该载体可以互补E1A和E1B中的缺失。通过为这种载体提供编码腺病毒E4orf6的核酸,当在293或911细胞上生长时,在不存在额外的野生型污染问题的情况中,可以有效进行噬斑纯化及良好生产。当然,在PER.C6中也可以通过其它方式防止野生型腺病毒污染。
本发明的重组载体的另一个优点是不需要从整合进基因组的核酸中产生特殊的细胞系腺病毒E4-orf6。尽管存在这类细胞系,但如扩大生产等生产参数和/或调节问题的解决还未达到如PER.C6TM细胞系相同的程度。这至少部分是由于越来越多的外源基因插入细胞系的基因组中,难以保持所有外源序列的稳定性(或其表达)所致。在本发明中,发现至少一些与基于非腺病毒血清型5的载体的低产量及来自亚群B如腺病毒血清型7、11和35的腺病毒血清型的载体在腺病毒血清型5包装细胞上的稳定性相关的问题可以用本发明的重组腺病毒载体克服。
应清楚本发明的两个主要方面可以组合以提供可在易于利用的常规包装细胞上生长的稳定的重组腺病毒。本发明因此提供了包含E1区中有至少一个缺失的重组核酸分子的一种重组腺病毒,特征在于所述重组核酸分子中存在编码提高pIX基因表达的E1B 55K基因产物的序列的至少一部分,附带条件是所述重组核酸分子不编码功能性E1B 55K基因产物;所述重组腺病毒进一步包含第一种血清型腺病毒的结构和非结构元件,其中所述腺病毒进一步包含编码功能性E4-orf6蛋白或其功能部分、衍生物和/或类似物的一个序列,其中所述序列是衍生自与所述第一种血清型不同的第二种血清型的腺病毒的E4-orf6编码序列。另外或者除了具有至少一部分E1B 55K序列之外,所述核酸可具有由一个异源启动子调节的一个pIX基因产物。优选地,所述第二种血清型及因此的E4-orf6序列衍生自C群腺病毒,更优选衍生自腺病毒血清型5。优选地,所述第一种血清型来自C群之外的一个亚群,优选亚群B血清型如Ad11、Ad14、Ad16、Ad21、Ad34、Ad35或Ad50。在一个实施方案中,重组腺病毒进一步包含编码非腺病毒蛋白、多肽或肽的一个序列。当重组腺病毒中的E4-orf6与Ad5 E1基因产物相容时,例如当所述腺病毒中的E4-orf6衍生自Ad5时,这种重组腺病毒是稳定的并可以在易于利用的包装细胞上生长,所述包装细胞如含有Ad5E1的包装细胞,优选PER.C6TM细胞。防止能复制的腺病毒或含有功能性E1蛋白的腺病毒颗粒产生是本领域中意识到的一个问题,并且已经通过防止腺病毒中存在的E1序列与包装细胞中存在的序列重叠而解决(US patent 5,994,128)。在本发明的实施方案中,例如包含通过影响pIX表达而提高病毒稳定性的E1B55K序列的Ad35衍生的腺病毒,组合包含来自Ad5的E1区的包装细胞,不导致伴随包含编码功能性EIB 55K蛋白的核酸的重组腺病毒伴随形成的同源重组。因此本发明的另一方面提供了一种包装系统,其包含一种包装细胞和一种包含第一种血清型的腺病毒的结构和非结构元件的重组腺病毒,其中所述包装细胞表达衍生自第二种血清型的腺病毒的编码至少E1B-55K蛋白或其功能部分、衍生物和/或类似物的核酸,其中所述重组腺病毒可以在所述包装细胞中复制以产生稳定的重组腺病毒,所述腺病毒包含在E1区有一个缺失的核酸分子并进一步包含一部分编码E1B-55K基因产物的序列,所述核酸进一步包含编码与所述包装细胞中的所述可表达的E1B-55K蛋白相容的功能性E4-orf6蛋白或其功能部分、衍生物和/或类似物的序列。优选所述第一种血清型是Ad 35或Ad11。优选所述第二种血清型是Ad5。更优选地,所述包装细胞是PER.C6TM。优选地,编码功能性E4-orf6蛋白或其功能部分、衍生物和/或类似物的所述序列衍生自第一种血清型的腺病毒。这个系统可以使重组腺病毒复制而不伴随含有功能性E1蛋白的腺病毒产生。或者,所述核酸可以含有在外源启动子控制下的pIX编码序列。本发明的另一方面是提供一种生产稳定的包含第一种血清型腺病毒的结构和非结构元件的重组腺病毒的方法,其中所述重组腺病毒包含衍生自其核酸分子在E1区具有缺失的腺病毒的重组核酸分子,并包含衍生自至少一部分编码提高pIX蛋白表达但不引起来自所述核酸分子的功能性E1B-55K蛋白表达的E1B-55K基因产物的序列和/或具有在异源启动子控制下的pIX编码序列,所述方法包含如下步骤:a)将腺病毒的必需元件提供给可表达形式的衍生自第二种血清型腺病毒的E1B-55K编码序列或其功能部分、衍生物和/或类似物的互补细胞,以使得可以通过所述互补细胞装配所述重组腺病毒载体,其中所述元件包含与所述第二种血清型不同的第一种血清型腺病毒的至少一些结构和非结构元件及编码与所述互补细胞中所述可表达的E1B-55K蛋白相容的功能性E4-orf6蛋白或其功能部分、衍生物和/或类似物的一个序列;b)在生产和装配重组腺病毒的条件下在培养基中培养所述互补细胞;及c)从所述培养基和/或互补细胞中收获如此生产的重组腺病毒。本发明进一步提供了本发明的重组腺病毒在治疗人或动物、免疫接种、基因治疗方面及制备治疗疾病或失调的药物中的应用方法。本发明还提供了包含本发明的腺病毒的药物制品。
现在通过一些实施例例证本发明,这些实施例非限制本发明的范围。
实施例
除非特别说明均使用标准分子生物学方法(例如Sambrook andRussel,2001)。引物序列在表IV中提供。
实施例1:针对E1缺失的Ad35病毒的基于PER.C6TM的互补细胞系在补加最多10%的FBS(Gibco BRL)和10mMMgCl2(4.9M原液,Sigma))的PER.C6培养基(DMEM)(Gibco BRL)中,将PER.C6细胞以3×106个细胞/皿的密度接种于10cm培养皿中。两天后,将9个培养皿用1μgScaI线性化的pIG35.55K DNA(如前述)转染及将9个培养皿用1.5μgScal线性化的pIG35.55K DNA转染。单独的对照培养皿用1或1.5μg ScaI线性化的pAdApt35.LacZ(WO00/70071所述)转染以监测转染效力并用1或1.5μg ScaI线性化的pcDNA.nlsLacZ转染。pcDNA.nlsLacZ(W099/55132所述)是一种基于pcDNA3的质粒(Invitrogen),具有由CMV启动子驱动的nlsLacZ基因。pcDNA.nlsLacZ还含有一个neor表达盒。作为阴性对照,将一个额外的培养皿用线性化的pAdApt35.LacZ转染,这是缺少neor选择基因的一种构建体。所有转染均用LipofectAmine转染试剂盒(Invitrogen/Life Technologies)根据厂商说明书使用5ml LipofectAmine试剂/μgDNA进行。将细胞用所述转染混合物温育4小时,之后将该培养基用PER.C6培养基更换。第2天,除了用1或1.5μgpAdApt35.LacZ转染的两个培养皿之外,将培养基用含有0.5mg/mlG418(Gibco BRL)的培养基更换。前者用于监测在转染两天后的LacZ表达。在将这些培养物经X-gal染色后,在用1.5μgDNA转染的培养皿中在略微更蓝的细胞为大约40%时评估转染效率。在剩余的转染的培养皿中每周更新两次选择培养基。在第一次加入选择培养基之后的两周内,阴性对照培养皿(用1.5μgpAdApt35.LacZ转染)中大多数细胞死亡。在用pcDNA.nlsLacZ转染的培养皿中,可见明显的细胞克隆。由于用pIG35.55K转染的细胞似乎对G418更具抗性,因此在转染后3周将浓度提升至0.75mg/ml。在3和7天后,从用pIG35.55K转染的培养皿中挑取共196个细胞克隆并接种于96孔平板的各个孔中。
将从两个用1μgpIG35.55K DNA转染的10cm培养皿中挑取集落后剩余的细胞经胰蛋白酶消化,集合(pooled)并扩展以提供集合(pool)PER55K(1.0)。对用1.5μg转染的两个培养皿同样处理。将PER55K(1.0)细胞集合扩展并以3.5×106个细胞/瓶的密度接种于4个T25培养瓶中,进行转染以测试病毒产生。另外,以相同密度将亲代PER.C6细胞接种于3个T25培养瓶中。将pAdApt35.eGFP(一种基于pAdApt35IP1(WO00/70071中所述)的衔接质粒,但也含有作为衍生自pIPspAdapt.eGFP的HindIII-BamHI片段克隆进pAdApt35IP1中的绿色荧光蛋白作为标记基因(WO02/24933中所述))用PacI消化以从质粒主链中释放腺病毒序列。将pWE.Ad35.pIX-rITR(WO00/70071中所述)用NotI消化以从粘粒主链中释放腺病毒序列。将具有PER.C6细胞的2个培养瓶和具有PER55K(1.0)细胞的2个培养瓶均用2μg消化的pAdApt35.eGFP和6μg消化的pWE.Ad35.pIX-rITR转染。将每种细胞系的1个培养瓶用8μgpAdApt35.LacZ转染以监测转染效力。具有PER55K(1.0)细胞的剩余的培养瓶作为阴性对照并如其它培养瓶同样处理,但不接受转染混合物。所有转染均用LipofectAmine(Invitrogen/Life Techn.)根据厂商说明书使用针对每次转染的共8μgDNA和40μlLipofectAmine试剂。在温育4小时后将转染混合物除去并加入新鲜培养基。在接种细胞后一天进行转染,再两天后将T25培养瓶中的细胞移至一个T80培养瓶中,LacZ对照转染除外。在温和固定(mild fixation)之后用X-gal溶液染色。用染色溶液温育5小时后,在这两个培养瓶中估计蓝色细胞的百分比为大约90%,示出这两种细胞系均良好转染。在移至T80培养瓶中4天后,转染的PER55K(1.0)培养物示出起始CPE(致细胞病变作用,表明病毒复制)为接近100事件(events)/培养瓶。将未转染的PER55K(1.0)细胞生长至铺满,无CPE迹象。在转染的PER.C6培养物中,在铺满的细胞单层中仅观测到三个CPE事件。再三天后,转染的PER55K(1.0)培养物示出完全的CPF,所有细胞均是圆形的并成块分散。相反,在PER.C6培养物中,很少量CPE不进展,所有细胞仍是单层。这证实了早期的观测结果,即E1缺失的基于Ad35的病毒的产生对PER.C6非常无效。正如所期望的,未转染的PER55K(1.0)培养物示出无CPE的一铺满单层。收获具有完全CPE的PER55K(1.0)培养瓶中的细胞和培养基并进行两次冷冻/解冻循环,之后在台式离心机中在3000rpm离心10分钟除去细胞碎片。所得粗制裂解物之一用于感染T175培养瓶中PER55K(1.0)细胞的新鲜培养物(1.5ml/瓶)。4天后在完全CPE时收获细胞和培养基。这示出感染的病毒已经在初始转染中形成。GFP表达通过荧光显微镜检测粗制裂解物感染的A549细胞而证实。然后如下所述使用该粗制裂解物分析各个克隆中E1缺失的Ad35.AdApt.eGFP病毒的互补。
将从pIG35.55K转染的PER.C6细胞中挑取的前述克隆扩展并测试其维持Ad35.AdApt.eGFP复制的能力。为此将所述克隆以两种密度接种于6孔平板中,1天后用15ml上述粗制裂解物感染。随后监测CPE。以此方式测试的146个克隆中,19个克隆在第2或3天出现完全CPE,68个克隆在第5或6天出现完全CPE。剩余的克隆仅呈现部分CPE或者示出少量非进展性事件。后者从作为阴性对照的PER.C6细胞中不能辨别。
基于这些结果,选择24个克隆,在用pAdApt35.GFP衔接质粒和大pWE.Ad35.pIX-rITR粘粒克隆转染后对其进一步筛选产生重组E1缺失的病毒的能力。为此将克隆铺板于T25培养瓶中并用2μg的衔接质粒和6μg的主链质粒使用上述LipofectAmine进行转染。在转染2天后,将细胞移至T80培养瓶中以防止培养物过度铺满。在24个克隆中,在经过移至T80后3天有5个克隆出现完全CPE,另有13个克隆随后出现完全CPE。剩余的6个克隆示出无CPE或者仅开始CPE。对比之下,在移至T80培养瓶之后4-6天,PER.C6细胞上的E1缺失的Ad5载体的常规产生通常导致完全CPE。
这示出新克隆有效互补E1缺失的腺病毒载体。上述克隆(16个克隆)之一用于产生及生产多批次的含有不同转基因的E1和E1/E3缺失的Ad35病毒。为此,将得自如上述转染但使用不同衔接质粒的粗制裂解物中的病毒在新细胞系中进行噬斑纯化。对单一噬斑测试转基因活性,然后在4-8个三层培养瓶(3×175cm/瓶)中扩大培养规模生产。在完全CPE时收获细胞并如针对腺病毒同样常规将病毒释放并纯化。病毒颗粒的量通过HPLC(Shabram et al.,1997)确定。表I示出在具有PER55K克隆#16细胞的三层培养瓶上E1和E1/E3缺失的Ad35病毒的中等规模生产的下游处理(downstream processing)后的产量。纯化的病毒颗粒的量与在PER.C6细胞上基于Ad5的载体的产量相似。
我们推断已经产生了有效互补完全E1缺失的基于Ad35的载体的多个细胞系。因此,Ad5互补细胞系中Ad35 E1B-55K表达促进Ad35载体的复制。
实施例2:pWE.Ad.pIX-rITRΔE3的产生
人腺病毒的早期区-3含有干扰宿主对腺病毒感染的免疫应答的蛋白质的多个编码区。当腺病毒载体用作疫苗载体时,这种干扰是不希望的。因此,我们构建了一种缺少E3区的Ad35主链粘粒。
为此,将构建体pBr.Ad35.PRn(图15;EP1054064中实施例13所述)用StuI和MluI消化并使用琼脂糖酶(Roche)根据厂商说明书将17.3kb载体片段从低解链点(LMP)凝胶中纯化。接着,使用引物35E3for和35E3rev在pBr.Ad35.PRn上产生PCR片段。为进行扩增,根据厂商说明书使用Pwo DNA聚合酶(Roche)进行,程序设定为:94℃2分钟,30次循环(94℃30秒,58℃30秒及72℃1分钟)及最后在68℃温育8分钟。将833bp PCR产物使用QIAquick PCR纯化试剂盒(Qiagen)纯化并用MluI和StuI消化。消化的DNA使用QIAquick凝胶提取试剂盒(Qiagen)从凝胶中纯化。连接这两个分离的片段并转化进DH5α感受态细胞(Invitrogen/Life Tectnologies)中以产生pBr.Ad35.PRnΔE3(图25)。该质粒通过限制分析进行检测并对PCR扩增的插入体进行测序。然后将E3缺失克隆进pWE.Ad35.pIX-rITR粘粒主链中。
为此,将pWE.Ad35.pIX-rITR用PacI消化并将DNA通过用异丙醇沉淀而纯化并用70%EtOH洗涤。在milliQ水中再悬浮之后,将该DNA用SwaI消化并将含有22.8kb载体的片段使用上述琼脂糖酶从LMP凝胶中纯化。将构建体pBr.Ad35.PRnΔE3以相同方式用PacI和SwaI消化并也使用琼脂糖酶分离16.6kb片段。将这两个分离的片段连接,每个片段使用0.5-0.6μg。然后将连接的片段使用λ-噬菌体包装提取物(Stratagene)根据厂商说明书进行包装并与STBL-2细胞混合。将细菌铺板于LB+Amp平板上并分析所得菌落中正确构建体的存在情况。这样提供了构建体pWE.Ad35.pIX-rITRΔE3(图1)。E3缺失从Ad35序列的第27648位核苷酸延伸至第30320位核苷酸(WO00/70071中所述)并因此跨越2.6kb的区域。
NotI消化的pWE.Ad35.pIX-rITRΔE3和pIPsp-1(New EnglandBiolabs)消化的pAdApt35.eGFP在PER55-克隆#16细胞(如前述)上的共转染产生表达GFP的基于Ad35的病毒。在从这些病毒中分离病毒DNA,E3区的PCR扩增示出正如所期望的该病毒缺失E3序列的2.6kb。
实施例3:包装E1缺失的基于Ad35的载体大小的限制
含有不同插入体的基于Ad35的E1缺失的和E1/E3缺失的载体通过用以下转染的PER55K-克隆#16细胞产生(见实施例1所述)
1.5μg的携带用PacI或pIPsp-1酶消化的特异性转基因的Ad35衔接质粒转染以从所述质粒载体序列中释放腺病毒插入体及,
4.5μg经NotI消化的pWE.Ad35.pIX-rITR或者用NotI酶消化的pWE.Ad35.pIX-rITRΔE3进行转染。
衔接质粒的右侧和主链质粒的左侧末端含有介导重组事件的同源序列,其产生完整的E1缺失的病毒基因组(如WO00/70071所述)。
针对每组构建体使用30μlLipofectamine试剂(Invitrogen/LifeTechnologies)根据厂商说明书进行转染。将转染混合物加入于T25培养瓶中70%铺满的PER55K克隆16细胞中。对以下组合进行转染:
1.pAdApt35IP1+pWE.Ad35.pIX-rITR
2.pAdApt35IP1+pWE.Ad35.pIX-rITRΔE3
3.pAdApt35eGFP+pWE.Ad35.pIX-rITR
4.pAdApt35eGFP+pWE.Ad35.pIX-rITRΔE3
5.pAdApt35Luc+pWE.Ad35.pIX-rITR
6.pAdApt35Luc+pWE.Ad35.pIX-rITRΔE3
7.pAdApt35LacZ+pWE.Ad35.pIX-rITR
8.pAdApt35LacZ+pWE.Ad35.pIX-rITRΔE3
将衔接质粒用pIPsp-1酶消化以从质粒载体主链中释放腺病毒序列。将pWE.Ad35.pIX-rITR和pWE.Ad35.pIX-rITRΔE3用NotI消化之后出于同样原因进行转染。衔接质粒及pWE.Ad35.pIX-rITR主链粘粒的产生如先前WO00/70071所述。pWE.Ad35.pIX-rITRΔE3的产生如前文所述。
在转染两天后,将细胞传代至T80并进一步温育直至获得完全CPE。在观测到完全CPE后收获细胞和培养基。将该混合物进行一次冷冻/解冻循环并在1500rpm旋转15分钟以沉淀细胞碎片,之后收集上清。将以此方式获得的粗制裂解物用于分离病毒DNA。为此,将275μl粗制裂解物与10μl的10mg/mlDNaseI在37℃温育30分钟。随后加入6.0μl 0.5M EDTA(pH8.0),7.5μl 20%SDS和1.5μl 20mg/ml蛋白酶K并涡旋混合。然后将此混合物在50℃温育1小时。最后,使用GeneClean Spin试剂盒(Bio 101,Inc.)分离病毒DNA。在20μlmilliQ H2O中洗脱病毒DNA之后,转基因区域通过PCR扩增进行分析。为此,使用引物AdApt35CMVF和35pIXR。使用Taq DNA聚合酶(Invitrogen)用2μl分离的病毒DNA进行扩增。反应混合物含有5μl10×缓冲液(Invitrogen),2μl 50mM MgCl2,5μl 2mM dNTPs,3μl每种引物(10μM原液)及2.5单位的Taq酶,总体积为50μl。程序设定为94℃2分钟,随后30次(94℃30秒,60℃30秒及72℃4分钟)循环。对照反应是对5ng衔接质粒进行。在完成PCR之后,将5μl反应物加样于凝胶上进行分析。图2示出了上述转染的结果。引物从CMV启动子的5’端至pIX编码区的5’端扩增序列。从图2中可以看出,没有转基因或具有GFP插入体的病毒示出期望的条带(与对照质粒相对比;针对每个病毒的泳道PL)。可见较小片段具有较大插入体,荧光素酶和LacZ,这些缺失在总长度较大的病毒中更明显(与有或无E3的LacZ或Luc病毒相对比)。因此,基因组长度的增加与转基因区域中缺失的出现相关。Ad35.AdApt.eGFP和Ad35.AdApt.LacZΔE3的总基因组长度分别为33.7和33.4kb(如图2所示),这个事实与缺失仅发现在LacZ病毒样品中的结果相似,表明前者E1区的序列或插入体的大小也可以影响缺失的出现。
实施例4:腺病毒血清型5和35的pIX基因区的序列对比
重组Ad35病毒的基因组长度增加在转基因区域中出现缺失的发现可以提示,依此类推在pIX-缺陷的Ad5病毒中(Ghosh-Choudhury etal.,1987),存在壳体稳定性的问题。可以产生总长度为36.7kb的Ad35.E1B+.AdApt.Luc病毒(其中E1A序列由AdApt.Luc盒置换)的事实表明E1B的缺失起作用。这也许是通过E1B蛋白之一的自身功能或者通过这个区域中未知的影响其它腺病毒蛋白表达的调节序列的功能所致。我们的知识还未描述E1B蛋白自身影响腺病毒的包装能力。然而,已经描述了E1B-21K蛋白非特异性使转染的DNA稳定(Herrman and Mathews,1989)及21K蛋白中的突变在感染期间导致细胞和病毒DNA降解(Pilder et al.,1984;White et al.,1984)。由于Ad5E1B-21K蛋白在PER.C6细胞中表达,因此这些发现就我们的观测结果未提供解释。
PIX基因就位于E1B-55K编码区的3’端。就Ad5而言,已知pIX启动子和编码序列位于E1B转录区内,因为pIX和E1B共有聚腺苷酸化信号。pIX表达所必需的最小启动子序列已经在Ad5的情况中加以研究(Babiss and Vales,1991)。示出含有上游Sp1位点和TATA框序列的启动子片段就pIX表达是足够的。Sp1位点与TATA框以及TATA框自身的序列之间的间隔示出影响pIX表达水平。Ad35中的相应区域是否对稳定病毒而言也足以驱动pIX表达至足够高的水平还未知。序列对比表明Sp1位点和TATA序列均与在Ad5pIX启动子中发现的那些序列不同。使用可从Genbank中获得的序列信息,对来自不同亚群的血清型的近端pIX上游序列(即E1B-55K的终止密码子与pIX基因的起始密码子之间)进行对比。以下具有SEQ ID NO:和Genbank参考序列的腺病毒用于进行对比:Ad2(SEQ ID NO:45;Genbank NC_001405),Ad5(SEQ ID NO:46;Genbank M73260),Ad12(SEQ ID NO:47;Genbank NC_001460),Ad9(SEQ ID NO:48;Genbank AF099665),Ad40(SEQ ID NO:49;Genbank L19443),Ad4(SEQ ID NO:50;NC_003266),Simian 25(SEQ ID NO:51;GenbankAF394196),Ad7(SEQ ID NO:54;GenbankAD7001)。Ad35序列(SEQID NO:52)在WO00/70071中公布。Ad11序列(SEQ ID NO:53)在先前未公布,在此提供。图3A示出上述序列在E1B-55K蛋白的终止密码子(全部序列中的前3个核苷酸)与pIX蛋白的起始密码子(最后3个核苷酸)之间的序列对比。Ad2和Ad5中的Sp1位点和TATA序列在方框中标示。在大多数情况中,在其它序列中针对Sp1和TATA框没有足够的同源性。因此,由Bucher,P(1990)公布的GC和TATA框的共有序列用于鉴别多种序列中推定的Sp1和TATA框。图3b示出推定的Sp1和TATA框序列及它们之间的间隔。分别属于亚群A、D和F的Ad12、Ad9和Ad40具有Sp1和TATA序列,其与共有序列相当匹配。然而,两个框之间的距离小于Ad5和Ad2之间的距离。这不是不同寻常的,因为Ad5E1B启动子也含有间隔11个核苷酸的Sp1框和TATA序列。然而,Sp1与TATA框之间的Ad5pIX启动子序列缺失(20个核苷酸中的)9个核苷酸降低pIX水平(Babiss and Vales,1991)。亚群B血清型Ad35、Ad11和Ad7以及亚群E病毒Ad4具有不同的TATA框序列及在推定的Sp1序列与TATA框之间具有不的同间隔。人腺病毒4型中近端pIX区与猿猴腺病毒25(CV68)中该区域相同,这是最近提议作为治疗性载体的一种血清型(Farina et al.,2001)。因此,对于稳定的壳体,基于非人腺病毒pIX表达的复制缺陷的载体也不是足够的。
有充分理由相信pIX表达在Ad35病毒和其它人及非人腺病毒中是以不同方式调节的,调节序列或者甚至启动子序列自身位于E1B序列的上游或者甚至更上游。或者,由于pIX表达是由E1A蛋白激活的,因此在存在属于相同血清型或亚群的E1A蛋白的情况下获得高水平的pIX表达是可能的。
我们测试了将内源近端pIX上游序列改变为异源启动子以提高载体中pIX表达是否产生更稳定的病毒及更好的包装能力(如前述)。或者,pIX功能也许通过包装细胞系反式(in trans)递送。作为一个非限制性实施例,我们描述了具有与在Ad5病毒中发现的相同的非内源近端pIX启动子的重组的基于Ad35的病毒,并示出这些病毒与未改变的重组载体相比具有更好的稳定性。
实施例5:具有Ad5pIX启动子的衔接质粒的产生
pAdApt535是一种Ad35衔接质粒,其具有部分Ad5pIX启动子序列但其它部分与Ad35衔接质粒pAdApt35IP1(见WO00/70071所述)相同。其构建如下所述:
用引物SV40for和pIX5Rmfe产生第一个PCR片段。该反应使用Pwo DNA聚合酶(Roche)根据厂商说明书进行,但最终混合物中具有3%DMSO。取pAdApt作为模板,这是Ad5 E1缺失的病毒的一种衔接质粒(100ng;见WO00/70071所述)。程序设定如下:94℃2分钟,然后进行30次(94℃30秒(解链),52℃30秒(退火)及72℃30秒(延伸))循环,随后72℃8分钟。所得PCR片段含有来自pAdApt的SV40聚腺苷酸化信号的3’端和Genbank登记号M73260所示从3511至3586位核苷酸的Ad5pIX启动子区域及在3’端的一个MfeI位点。
如上述产生第二个PCR片段,但使用引物pIX35Fmfe和35R4进行。取100ng pAdApt35IP1作为模板,PCR程序的退火设定在58℃30秒,延伸设定在72℃90秒。这个PCR扩增了从3467至4669位核苷酸的Ad35序列(序列编号如WO00/70071所述)并在5’端加入了一个MfeI位点。
然后将这两个PCR片段用MfeI消化并使用Qiagen PCR纯化试剂盒(Qiagen)根据厂商说明书进行纯化。纯化的片段的浓度通过将样品在琼脂糖凝胶上运行而评估,并将接近等摩尔量的两个片段混合于40μl体积的含有5μgDNA,4μl 10×连接酶缓冲液和2μl连接酶(NewEngland Biolabs)的连接反应物中。在室温温育2小时以上的时间后,将该混合物加样于TAE中的1.2%琼脂糖凝胶上,使用Geneclean II试剂盒(Bio101,Inc.)根据厂商说明书分离长度为1.4kb的DNA片段。
将该DNA在30μl无菌水中洗脱并将1μl用于PCR扩增反应中,使用如上述引物SV40for和35R4进行。如上述进行PCR,退火温度为52℃,延伸时间为90秒。所得产物使用Qiagen凝胶提取试剂盒从凝胶中分离并用AgeI和BglII消化。所得0.86kb条带使用GenecleanII试剂盒根据厂商说明书从凝胶中分离。
将pAdApt35.Luc(见WO00/70071所述)也用BglII和AgeI消化并将5.8kb载体片段使用Geneclean II试剂盒如上述从凝胶中分离。将这个片段如前述含有Ad5-Ad35嵌合pIX启动子的分离的BglII-AgeI片段连接,产生pAdApt535.Luc(图4)。
然后如下产生含有Ad5 pIX启动子的其它衔接质粒:
将pAdApt535.Luc用BglII和ApaI消化并使用Geneclean II试剂盒根据厂商说明书从凝胶中纯化1.2kb的插入体。将pAdApt35IP1也用BglII和ApaI消化并如上述分离3.6kb的载体片段。将这两个分离的片段连接产生pAdApt535(图5)。接着,使用标准克隆技术使用pAdApt535将其它标记基因如eGFP(衍生自pAdApt35.eGFP)和LacZ(衍生自pAdApt35.LacZ)克隆进多克隆位点,产生pAdApt535.eGFP和pAdApt535.LacZ。
实施例6:具有含有Ad5pIX启动子的衔接质粒的E1缺失的基于Ad35的载体的产生
重组病毒如上述通过在PER55K克隆16细胞上转染衔接质粒和Ad35载体主链粘粒而产生。为此,使用如下质粒:
T1.pAdApt535eGFP+pWE.Ad35.pIX-rITR
T2.pAdApt535eGFP+pWE.Ad35.pIX-rITRΔE3
T3.pAdApt35Luc+pWE.Ad35.pIX-rITR
T4.pAdApt535Luc+pWE.Ad35.pIX-rITR
T5.pAdApt535Luc+pWE.Ad35.pIX-rITRΔE3
T6.pAdApt535LacZ+pWE.Ad35.pIX-rITR
T7.pAdApt535LacZ+pWE.Ad35.pIX-rITRΔE3
T8.pAdApt35LacZ+pWE.Ad35.pIX-rITR
T9.pAdApt35LacZ+pWE.Ad35.pIX-rITRΔE3
将衔接质粒在转染之前用PacI消化,除了pAdApt535.Luc和pAdApt35.Luc用pIPsp-1酶消化及pWE.Ad35.pIX-rITR和pWE.Ad35.pIX-rITRΔE3用NotI消化。将2μg每种衔接质粒和6μg的主链DNA与40μl Lipofectamine(Invitrogen/Life Technologies)根据厂商说明书混合并与T25培养瓶中70%铺满的PER55K克隆16细胞温育。4小时后除去转染培养基并将细胞在37℃/10%CO2中进一步温育。在转染两天后,将细胞传代至T80培养瓶并对致细胞病变作用(CPE)的出现情况评分。5天后所有培养物均示出进展中的或完全的CPE,T6(无CPE)和T8(CPE事件)除外。再两天后,T6和T8示出开始CPE,所有其它培养物均呈现完全CPE。通过收集培养基和细胞收获所有培养物。将该混合物在-20℃贮存。将样品解冻,将该混合物在1500rpm旋动15分钟以沉淀细胞碎片,收集上清。在一些样品中(4种LacZ表达病毒,T6-T9),取2ml用于再次感染于T80培养瓶中80%铺满的PER55K克隆16细胞以进一步扩增病毒滴度。在进展的(T6+T8)或完全(T7+T9)CPE中收获细胞和培养基并如上述制备粗制裂解物。
以此方式获得的粗制裂解物用于分离病毒DNA。为此,将275μl粗制裂解物与10μl的10mg/ml DNaseI在37℃温育30分钟。随后加入6.0μl 0.5M EDTA(pH8.0),7.5μl 20%SDS和1.5μl 20mg/ml蛋白酶K并涡旋混合。然后将该混合物在50℃温育1小时。最后,使用GeneClean Spin试剂盒(Bio 101,Inc.)分离病毒DNA。将病毒DNA在50μl milliQ水中洗脱并将5μl样品用于分析转基因区域。应注意pWE.Ad35.pIX-rITR+/-E3主链粘粒不改变并因此仍含有Ad35pIX启动子。由于该启动子位于粘粒的5’端,因此认为重组事件产生野生型Ad35启动子的机会很少。然而,不排除产生仍含有Ad35 pIX启动子的病毒。因此,针对每个病毒制备物进行两次特异性PCR扩增:第一次用引物组1(Ad35特异的)进行:AdApt35CMVF和AdApt35pIXrev。这个PCR特异性扩增含有Ad35pIX启动子的病毒中的转基因区域。对5μl分离的病毒DNA样品用重组Taq聚合酶(Invitrogen)根据厂商说明书进行PCR反应,但反应中使用4mMMgCl2和4单位的Taq酶。PCR程序设定为在94℃2分钟,随后进行30次(94℃30秒、60℃30秒及72℃5分钟)循环,最后以68℃8分钟结束。
第2次用引物AdApt35CMVF和pIX5Rmfe进行并因此特异性扩增含有Ad5 pIX启动子的病毒中的转基因区域(引物组2)。
对5μl分离的病毒DNA使用Pwo DNA聚合酶(2.5单位/μl,Genaxis)于含有0.3μl每种引物(100μM原液)、5μl2mMdNTP混合物、5μl 10×完全缓冲液(包含Mg2+),1.5μl DMSO和0.8μl Pwo酶的50μl体积反应液中进行PCR扩增。PCR程序设定如下:94℃2分钟,随后进行30次(94℃30秒、60℃30秒及72℃5分钟)循环,最后以68℃8分钟结束。在PCR期间,加热和冷却坡度设为2℃/秒。然后,将5μl加样缓冲液加入样品中并将8μl混合物加样于凝胶上进行分析。
含有Ad5pIX启动子序列和eGFP转基因(转染T1和T2)的E1和E1/E3缺失的Ad35病毒具有期望高度的PCR扩增条带而无较短的片段。图6a示出对携带E1缺失的荧光素酶的病毒(转染T3和T4)进行PCR扩增的结果。泳道5-8是用这两个引物组对AdApt535Luc(泳道5和6)和AdApt35Luc质粒(泳道7和8)进行的对照PCR。泳道1-4是对病毒DNA分离物进行的PCR。引物组1(特异于Ad35pIX区域)扩增期望长度的一个条带并示出Ad35.AdApt35.Luc病毒上另外的较短片段(泳道4;与图2荧光素酶+E3相对比)。相反,当用AdApt535.Luc质粒产生病毒时,引物组2(特异于Ad5 pIX启动子)仅示出无缺失片段的期望长度的一个条带(泳道1)。由此推断Ad5 pIX启动子序列的插入提高了Ad35-E1缺失的病毒的稳定性和包装能力。图6b证实了携带LacZ作为转基因的Ad35 E1/E3缺失的病毒的这些结果。泳道1-4是对AdApt535.LacZ和AdApt35.LacZ质粒使用每个引物组进行的对照PCR。尤其使用引物组1见到一些背景条带1(泳道2和4),但对同源样品就每个引物组也见到期望高度的强特异性条带(泳道1和4)。在转染及如上述一次扩增循环之后分离病毒DNA。引人瞩目地,引物组2在Ad35.AdApt535.LacZ病毒上产生期望的片段而无缺失片段(泳道5和9),而含有Ad35pIX启动子序列的病毒样品明显示出除了正确长度的片段之外缺失的片段(在扩增后可见(泳道11)。
总而言之,这些结果示出将Ad35-pIX启动子序列取代为Ad5-pIX启动子序列提高了具有较大基因组的病毒中转基因区域的稳定。较强的启动子或者额外的启动子元件甚至可以增强这种作用。
实施例7:pWE.Ad35-3481的产生
如上所示,粘粒pWE.Ad35.pIX-rITR中的腺病毒插入体在其5’端含有Ad35pIX启动子。这可以导致Ad35pIX启动子再次插入用基于pAdApt535的衔接质粒产生的病毒中。因此,产生一种新的Ad35主链粘粒,其缺少pIX启动子序列。为此,使用pIXcosF-2和Adapt35-3引物产生一个PCR片段。使用Pwo DNA聚合酶(2.5单位/μl,Genaxis)在含有3μl每种引物(10μM原液),5μl 2mM dNTP混合物,5μl 10×完全缓冲液(包含Mg2+),1.5μl DMSO,0.5μlPwo酶和10ngpAdApt35IP1模板的50μl体积反应液中进行扩增。PCR程序设定为94℃2分钟,随后进行5次(94℃30秒,58℃30秒及72℃1.5分钟)循环,然后进行25次(94℃30秒,60℃30秒及72℃1.5分钟)循环,最后在68℃8分钟结束。所得1.2kbPCR产物含有从第3481-4663位核苷酸的Ad35序列(根据WO00/70071公布的Ad35序列编号),AatII和NotI位点附于5’端。将PCR产物使用PCR纯化试剂盒(Qiagen)根据厂商说明书进行纯化并根据厂商说明书克隆进pPCR-Script Amp载体(Stratagene)中。然后将克隆的片段的序列通过测序证实,随后通过用AatII和AgeI消化从该构建体中除去。所得780bp片段使用Geneclean spin试剂盒(Bio101,Inc.)根据厂商说明书从凝胶中纯化。
将构建体pWE.Ad35ΔNdeI(如前所述)也用AatII和AgeI消化,所得12kb载体片段使用Geneclean spin试剂盒(Bio101,Inc.)根据厂商说明书从凝胶中分离。将这两个分离的片段连接产生构建体pWE.Ad35-3481ΔN deI。
构建体pWE.Ad35ΔNdeI的构建如WO00/70071所述,其含有从第3401位核苷酸至在第6541位核苷酸的NdeI位点的Ad35序列及从在第33167位核苷酸的NdeI位点至右侧ITR末端的Ad35序列,这两个Ad35片段通过NdeI位点连接(也见WO00/70071中图13所示)。
然后将pWE.Ad35-3481ΔNdeI用NdeI线性化,用CIP酶(NewEngland Biolabs)去磷酸化及使用Geneclean spin试剂盒(Bio101,Inc.)根据厂商说明书从凝胶中纯化。然后将这个载体片段与分离自Ad35wtDNA的26.6kbNdeI片段连接,之后将该混合物使用λ噬菌体包装提取物(Stratagene)根据厂商说明书用于包装粘粒。所得混合物用于转化STBL-2细菌(Invitrogen),产生pWE.Ad35-3481。
实施例8:pIG35.55K的构建
构建体pIG35.55K含有可操纵地与人磷酸甘油酸激酶启动子(hPGK)连接的Ad35E1B-55K基因的编码序列和HBV聚腺苷酸化序列。另外,其含有可操纵地与RSV启动子连接的新霉素抗性基因和HBVpA。pIG35.55K的构建如下所述。
将构建体pIG270(WO00/70071所述)用EcoRI消化,用Klenow酶处理并使用PCR纯化试剂盒(Qiagen)根据厂商说明书进行纯化。然后将回收的DNA用AgeI消化并将~5kb的载体片段使用Geneclean试剂盒(Bio101,Inc.)根据厂商说明书从凝胶中分离。接着,将Ad35E1B-55K序列使用35D21和35B3引物在pIG270模板DNA上通过PCR扩增。使用Pwo DNA聚合酶(Roche)根据厂商说明书对2ng模板DNA进行PCR扩增,但使用于PCR混合物中终浓度为3%的DMSO。程序设定为:94℃2分钟,随后进行25次(94℃30秒,56℃30秒及72℃30秒)循环,最后在72℃温育10分钟结束。所得PCR片段使用PCR纯化试剂盒(Qiagen)纯化并用NcoI消化。在经Klenow处理以补平突出端之后,将DNA用AgeI进一步消化并再次柱纯化。然后将如此处理的PCR片段克隆进上述制备的EcoRI/AgeI消化的载体片段中以产生pIG270.ΔE1AΔ21K。将pIG270.ΔE1AΔ21K用AvrII和XbaI消化并使用Klenow酶补平突出端。如上述将含有PGK启动子和Ad35E1B-55K的2.9kb片段从凝胶中分离。接着,将pRSVneo4(如前述构建)用BglII消化,用Klenow酶产生平端,去磷酸化并从凝胶中分离。然后将来自pIG270.ΔE1AΔ21K的平端的AvrII/XbaI片段与上述制备的pRSVneo4载体片段连接以产生pIG35.55K。
pRSVneo4如下产生:将构建体pRSVhbvNeo(WO00/70071所述)用ScaI和BamHI消化并将突出端用Klenow酶补平。将含有部分氨苄青霉素基因和RSV启动子的1070bp片段使用Geneclean试剂盒(BIO 101,Inc.)从凝胶中分离。接着,将pRSVhbvNeo用ScaI和EcoRI消化,用Klenow产生平端并如上述分离含有neo基因、HBVpA、载体和部分氨苄青霉素基因的3.2kb片段。然后将这两个片段连接产生pRSVneo4。
实施例9:由RSV启动子介导的提高的pIX表达增加Ad35病毒的稳定性
作为驱动pIX基因表达的一个异源启动子的实例,将RSV启动子插入含有LacZ或荧光素酶报道基因的Ad35衔接质粒中。RSV启动子相应于得自pRc-RSV(Invitrogen)的NruI/ApaLI片段。突出端使用Klenow酶(New England Biolabs)根据厂商说明书补平。将含有RSV启动子的388bp片段使用QIAquick凝胶提取试剂盒(Qiagen)从琼脂糖凝胶中分离。将衔接质粒pAdApt35.Luc和pAdApt35.LacZ用BglII线性化,随后经Klenow处理以产生平端。BglII消化正好在转基因表达盒的SV40聚腺苷酸化序列之后。关于基于pAdApt35的衔接质粒的描述见WO00/70071所述。然后将处理的衔接质粒使用虾碱性磷酸酶(SAP)根据厂商(Roche)说明书去磷酸化。分离的RSV启动子片段然后与每种处理的载体连接并转化入DH5α感受态细菌(Invitrogen)中。对菌落分析相对于pIX基因RSV启动子正向插入产生的pAdApt35.Luc.rsv和pAdApt35.LacZ.rsv。
另外,从通过PCR从缺失转基因区域的Ad35重组病毒中分离的序列中产生一个衔接质粒。对得自pAdApt35.LacZ和pWE.Ad35.pIX-rITR Ad35主链构建体的转染的粗制裂解物进行分析及随后的噬斑纯化示出该病毒在大约2.8kb的转基因区域中有一个缺失。将这个病毒的5’序列使用引物35F1和35R4从分离的DNA中经PCR扩增。该反应使用Pwo聚合酶(Roche)根据厂商说明书进行。程序设定如下:94℃2分钟,然后进行5次(94℃30秒;48℃30秒;72℃2.5分钟)循环,随后进行25次(94℃30秒;56℃30秒;72℃2.5分钟)循环,最后在68℃8分钟结束。
所得2kb片段通过PCR纯化试剂盒(Qiagen)纯化并根据厂商说明书克隆进pCR-Script-Amp载体(Stratagene)中,产生pCR.Ad35Δ2.8kb。对这个质粒进行测序以确定缺失程度。这种缺失影响大多数CMV启动子、转基因和SV40polyA。这导致CMV启动子5′的317bp与pIX基因上游的Ad35序列连接。这个CMV片段含有3个GC-框和21-bp重复(Boshart et al.,1985)。可能,CMV启动子的剩余序列可以增加pIX表达,产生更稳定的病毒。另一种可能性是病毒较小则导致稳定性增加。为对此进行研究,将一个完整表达盒以如下方式克隆回来,产生具有这个新的衔接质粒的病毒。含有扩增的序列的pCR-Script-based载体(更名为pCR.C4)在ΔCMV-pIX序列之前有一个唯一的AvrII位点。如上述将该载体用AvrII线性化,用Klenow酶产生平端并使用SAP酶(Roche)去磷酸化。将衔接质粒pAdApt535.LacZ(实施例5)和pAdApt.Luc(WO00/70071)用AvrII和BglII消化并将DNA用Klenow处理以补平突出端。将相应于LacZ和荧光素酶表达盒(CMV-TG-pA)的片段如上述从凝胶中分离并与AvrII线性化的pCR.C4载体连接。如上述转化感受态细胞并选择所述表达盒与左侧ITR正向的菌落,产生pCR.C4.LacZ和pCR.C4.Luc。
使用以下新的衔接质粒如实施例6所述产生Ad35病毒:用PacI消化的pCR.C4.LacZ,用ApaI消化的pCR.C4.Luc及用PI-PspI消化的pAdApt35.LacZ.rsv和pAdApt35.Luc.Rsv。
将该衔接质粒在PER55K细胞(WO02/40665)上与用NotI消化的pWE.Ad35.pIX-rITR或pWE.Ad35.pIX-rITRΔE3共转染。另外,将衔接质粒pBr.Ad35.ΔE1AΔ21K.Luc(如下述构建)用PI-PspI消化并与NotI消化的pWE.Ad35.pIX-rITR共转染。在完全致细胞病变作用(CPE)时,通过一次冷冻/解冻循环收获培养物并离心以除去细胞碎片。然后在将PER55K细胞接种于T80培养瓶中的前一天将300μl的所得澄清的裂解物用于再次感染PER55K细胞。在完全CPE时,制备粗制裂解物并用于感染A549细胞以测试转基因表达并对PER55K细胞进行噬斑分析。
将A549细胞以5×105个细胞/孔的密度接种于6孔平板中,5小时后用10、1或0.1μl每种LacZ病毒原液感染并温育两天。然后将A549细胞针对LacZ活性进行染色并计数蓝色细胞。蓝色细胞的百分比示于表II。
当RSV启动子驱动pIX基因表达时,与缺失的CMV启动子相比LacZ表达病毒显然更稳定。这些结果使用荧光素酶病毒证实。为测定荧光素酶病毒活性,将A549细胞以1×105个细胞/孔的密度接种于24孔平板中,并用10、1、0.1或0.01μl的病毒原液感染并温育。在两天后,将细胞用PBS洗涤两次及再悬浮于100μl裂解缓冲液(Promega)中,在-20℃贮存直至使用。使用Steady-Glo荧光素酶分析系统(Promega)根据厂商说明书测定荧光素酶。结果示于表III。
在实施例3中我们描述了含有E3区和AdApt.Luc或AdApt.LacZ表达盒的完全缺失E1的Ad35病毒是不稳定的。显然,在新描述的构建体中,在pIX前面的缺失的CMV启动子不阻止转基因区域的缺失。然而,使用驱动pIX基因的RSV启动子,我们现在能产生比野生型更长的病毒。Ad35.AdApt.Luc.rsv和Ad35.AdApt.LacZ.rsv分别为35kb和36.5kb(也见图2)。Ad35.ΔE1AΔ21K.Luc(36.4kb)也示出高度转基因活性。
我们接着测试了在噬斑纯化之后病毒是否是完整的。为此,将PER55K细胞以0.9×106个细胞/孔的密度接种于6孔平板中并用Ad35.AdApt.LacZ粗制裂解物的不同的10倍稀释液感染。将从10-5至10-8的稀释液铺板并在第2天加入一层琼脂覆盖。为此,将细胞首先用PBS洗涤,然后加入3ml预热的琼脂溶液,该溶液通过将2×MEM(GibcoBRL;9.14ml),FBS(Gibco;0.36ml),MgCl2(4.9M;0.037ml)与琼脂糖(SeaPlaque GTG;于水中2.5%,7.2ml)混合而制备。在凝固之后,将平板在37℃/5%CO2中进一步温育。4天后见到噬斑并将LacZ染色溶液加入孔中琼脂之上并使其排干。所有病毒均示出范围在10-7至10-9的清晰的单独的噬斑,但仅在具有驱动pIX基因的RSV启动子病毒的情况中,所有噬斑均染成蓝色。在缺失的CMV启动子驱动pIX的两种情况中,至少部分噬斑不染色。这清晰地示出可包装的基因组大小/稳定在具有调节pIX的RSV启动子的病毒中增加。
本发明首次提供了衍生自或基于腺病毒血清型5的缺少功能性E1B基因表达的一种稳定的重组腺病毒。这种腺病毒在E1区有至少一个缺失。在本发明提供的特殊实施方案中,使用本发明的方法,所述稳定的重组腺病毒具有4.2kb以上的外源插入序列及33.4kb以上的一个包装的基因组。因此本发明的一个方面是提供了一种稳定的重组腺病毒,其:a)携带4.2kb以上的一个外源核酸序列,和/或b)具有33.4kb以上的一个包装的基因组。在特殊的实施方案中,本发明提供了基于Ad35或Ad11的重组腺病毒,其:a)携带至少4.6kb的一个外源核酸序列,和/或b)具有33.8kb以上的一个包装的基因组。或者,所述包装的基因组大小分别为至少34.6、35.0、36.1和36.5kb。这些实施方案中的外源序列可包括驱动pIX表达的一个异源启动子。另一方面,所述稳定的重组腺病毒是血清型11。根据本发明这个方面的稳定的腺病毒可以在包装细胞上传代以提供一批重组腺病毒,10%以下,优选5%以下,优选无一单独的克隆在所述重组腺病毒中的外源序列中产生缺失,这例如可以通过实施例3中例证的PCR方法测定。
pBr.Ad35.ΔE1AΔ21K.Luc(见上文)如下产生。将构建体pBr.Ad35.ΔE1AΔ21K(WO02/40665)用HpaI消化,用CIP(NewEngland Biolabs)去磷酸化并从凝胶中分离5kb的载体片段。将构建体pBr.Ad35.ΔE1A.Luc也用HpaI消化并从凝胶中分离3.3kb的插入体,并与分离的载体片段连接。在转化入感受态STBL-2细胞(Invitrogen)之后,选择具有正确方向的插入体的菌落。这样产生了构建体pBr.Ad35.ΔE1AΔ21K.Luc。pBr.Ad35.ΔE1A.Luc(也称为pBr.Ad35.E1B+.Luc,因为其仍含有E1B区)通过将取自经AvrII和BglII消化及用Klenow酶产生平端的pAdApt.Luc的AdApt.Luc盒插入经SnaBI和HindIII消化及用Klenow产生平端的载体片段pBr.Ad35.leftITR-pIX(WO02/40665)中而产生。选择具有正向表达盒的集落,产生pBr.Ad35.ΔE1A.Luc。
实施例10:pIX调节序列的鉴别
先前的实施例示出其中E1A和E1B的编码区完全除去的Ad35重组病毒如果基因组大小增加则逐渐变得不稳定。我们在本申请中示出了加入驱动pIX表达的一个异源启动子可以克服这种不稳定性。在WO02/40665和前文中,我们揭示了保留完整E1B-55K编码序列的Ad35病毒可以在PER.C6上产生而且是稳定的。对保留全部E1B编码序列的病毒也同样(WO00/70071;Abrahamsen et al.,1997)。结合这些结果产生这样的可能性,即在亚群B病毒中pIX基因的表达与在亚群C中pIX基因的表达相比以不同方式调节。因为保留由E1B启动子驱动的E1B-55K基因的病毒是稳定的(见上文),因此pIX调节序列可能位于这个区域中。为对此进行研究,我们产生了保留不同长度的55K序列的3’端的一系列构建体。为此,首先如下产生pBr.Ad35ΔSM.AdAptLacZ。将构建体pBr.Ad35.1ITR-pIX(见WO00/70071中所述)用SnaBI和MfeI消化,用Klenow产生平端及用SAP酶(Roche)去磷酸化。然后将4.4kb的载体片段从琼脂糖凝胶中分离。将构建体pAdApt.LacZ(一种具有LacZ转基因插入体的基于Ad5的衔接质粒pAdApt;WO99/55132)用AvrII和BglII消化(及任选用SalI消化以增加片段大小的差别)及用Klenow酶产生平端。然后将4.2kbCMV.LacZ.pA插入体从凝胶中分离。然后将这两个分离的片段连接以产生pBr.Ad35.ΔSM.AdAptLacZ(图7)。方向可以通过限制性消化检测,因为以正确方向连接可恢复AvrII位点和MfeI位点。构建体pBr.Ad35ΔSM.AdAptLacZ在相对于pIX基因的野生型位置中保留0.6kb的3′ElB-55K序列(野生型Ad35第2804-3400位核苷酸)。先前我们已经示出这些55K序列不引起功能性E1B-55K蛋白的表达,因为在PER.C6细胞上增殖是不可能的(pBr.Ad35ΔSM;WO02/40665)。从pBr.Ad35ΔSM.AdAptLacZ开始,可以通过用MfeI(MunI的同切点酶)和或者StuI、NsiI或BglII消化,随后在5’或3’突出端分别使用Klenow或T4DNA聚合酶平端化突出末端而产生680bp(0.7kb)的E1B-55K区域的不同缺失。将消化的DNA再次连接产生功能性衔接质粒,其然后用于在PER55K细胞上通过与如上述pWE.Ad35.pIX-rITR共转染而产生重组病毒。通过使用酶DraIII、Bsu36I、BssHII或BamHI,通过本领域已知的方法对所述载体进行部分消化(LacZ基因也含有这些酶的一个识别位点),随后通过选择正确的克隆而产生另外的构建体。如上述针对Ad35.AdApt.LacZ.rsv构建体测试稳定性。稳定的构建体(即在转基因区域中不获得缺失)含有pIX表达的适当调节区域。另外,可以通过插入报道基因上游序列而直接测试给定序列中的启动子活性。pGL3basic(Promega)是这样的一种报道基因构建体。使用引物组Ad3555KmfeF和Ad35pIXNcoR扩增MunI与pIX基因起始处之间的区域。这个PCR(94℃2分钟;然后进行30次(94℃30秒,59℃30秒,72℃60秒)循环;随后68℃8分钟;根据厂商说明书使用Pwo(Genexis),另外具有3%DMSO)扩增了从2804至3491位的Ad35序列(编号如在野生型Ad35中),从而将pIX的起始密码子周围的序列改变为NcoI位点并在5’端导入一个HindIII位点。将这个扩增的片段用HindIII和NcoI消化并克隆进用相同的酶消化的pGL3basic中,产生pGL3-MN。然后pGL3-MN用于通过组合HindIII与例如PacI、NsiI、StuI、Bsu36I、BssHII或BglII消化,随后通过平端化突出末端和再连接而缺失荧光素酶编码区上游的序列。启动子活性通过使用lipofectamine试剂根据厂商说明书将获得的构建体瞬时转染进PER.C6细胞中而测试。在转染两天后,使用Steady-Glo荧光素酶分析系统(Promega)根据厂商说明书分析荧光素酶活性。或者,在pGL3basic载体中插入不同的区域,该区域是通过使用Ad35 E1B-55K区域中一特异性序列的5’(正向)引物并组合了Nco-pIXrev引物使一个HindIII位点附于5’端的PCR扩增而产生的。在此方式中,非限于存在进行克隆的一个唯一的限制位点。
PIX启动子的位置通过使用软件进一步研究以发现推定的启动子序列(Reese and Eeckman,1995)。图8示出了与55K编码序列(如在pBr.Ad35.ΔE1AΔ21K中)直接关联的E1B启动子的启动子分值(最小值设定为0.65)。标记为A的区域相应于E1B启动子,区域B和C位于55K编码区内。pIX上游区域不认为是启动子序列。在这三个区域中,区域C具有最高分值(0.96)(甚至高于已知的E1B启动子)因而可能包含影响pIX表达的序列。
为通过实验定位和鉴别一个可能的pIX启动子,产生一系列相应于55K编码序列的3’端的不同(重叠)部分的小片段。图9示出了这些片段及其相对于推定的启动子区域和pIX基因的位置。所述片段通过使用指定的酶进行限制性消化而产生。片段用Klenow(5’突出端)或T4DNA聚合酶(3’突出端)平端化并克隆进也经Klenow处理而平端化并经SAP处理(Roche)而去磷酸化的pGL3basic载体(Promega)的NcoI位点中。在转化感受态细菌中之后,通过限制性消化对获得的质粒检测插入体的方向。然后通过使用lipofectamine试剂根据厂商说明书将获得的荧光素酶构建体瞬时转染进PER.C6细胞中而分析启动子活性。空的pGL3basic质粒作为阴性对照。另外的对照通过将i)来自含有Ad5 pIX启动子的pAdApt535的BglII-MfeI片段,ii)一个388bp的NruI-ApaLI RSV启动子片段(如上述),或者iii)Ad35 pIX上游区域作为PCR片段克隆进上述pGL3basic的平端NcoI位点而产生。使用引物SV40-for及5′-磷酸化的Ad35pIXrev在pAdApt35IP1上扩增Ad35上游pIX区域。在扩增之后,将该DNA用BglII消化并用Klenow处理。
将构建体也转染进不含有腺病毒E1的人细胞(例如A549,Hela)中以研究E1A表达的依赖性。
将片段也克隆进衔接质粒pBr.Ad35ΔSM.AdAptLacZ(见上述)中以能产生重组病毒并研究病毒基因组的稳定性。为此,将构建体pBr.Ad35ΔSM.AdAptLacZ用MfeI和BglII消化,用Klenow酶平端化并去磷酸化。在从凝胶中分离所述载体片段后,可将DNA与上述片段连接(见图9)以产生具有不同长度的pIX基因上游55K片段的一组衔接质粒。病毒可以用上述构建体pWE.Ad35.pIX-rITR产生。用pBr.Ad35ΔSM.AdAptLacZ,pAdApt35.LacZ和pAdApt35.LacZ.rsv构建体进行对照转染。在出现完全CPE之时,通过一次冷冻/解冻循环收获细胞和培养基并用于再次感染新鲜的PER55K细胞。在完全CPE时再次收获细胞和培养基,制备粗制裂解物并用于进行噬斑分析。在出现噬斑后,加入X-gal染色溶液以检测LacZ表达。
上述实验的结果有助于发现Ad35E1B-55K区域中pIX调节序列的位置。
或者,pIX基因表达可以通过作为异源启动子的E1B启动子驱动以产生重组病毒。为此,将pAdApt535.LacZ用BglII和MfeI消化,随后经Klenow处理而平端化末端及去磷酸化。然后将如此处理的4.8kb载体片段从凝胶中分离。E1B启动子区域使用pBr.Ad35.leftITR-pIX作为靶DNA及Epr-F和Epr-R引物作为PCR片段而分离,而这两个引物均磷酸化。使用Pwo DNA聚合酶(Genaxis/Inno-train Diagnostik Gmbh)根据厂商说明书进行PCR。然后将所得151bp片段克隆进分离的载体中以产生pAdApt35Epr.LacZ。
该质粒然后用于产生基于Ad35的病毒并如前述测试稳定性。
为鉴别含有pIX序列的不同转录物,从感染的细胞中分离RNA并将含有pIX的RNA通过与标记的特异性探针杂交而鉴别。为此,将PER55K细胞用如下病毒以感染复数为10和50感染:wtAd35、Ad35.E1B.AdApt.Luc、Ad35ΔE3.AdApt.Luc、Ad35ΔE3.AdApt535.Luc、Ad35.AdApt.Luc.rsv。8小时后(wtAd35在2和18小时后)收获感染的细胞,使用TRI-zol试剂(Invitrogen)分离RNA。将该RNA在1.5%琼脂糖凝胶上进行大小分级分离,移至Northern印迹并与衍生自pIX编码区的32P标记的探针杂交。本领域已知该程序(见MolecularCloning:A laboratory manual,by Sambrook and Russell,2001或较早的版本)。如果包含已知RNA大小标记并给出含有pIX序列的RNA物种的指示,则RNA的长度可以确定。为鉴别在E1B启动子中开始的mRNA,可以将印迹剥离并再与5′21K探针杂交。不与E1B-21K探针杂交的含有pIX的转录物可能通过与E1B启动子不同的启动子产生。
由于可能的(及期望的)剪接事件,仍难以通过这种方法准确确定转录起始位点。这可如下实现。将分离的RNA逆转录为cDNA并使用GeneRacer系统(Invitrogen)根据厂商说明书使用pIX编码序列的反向引物将该cDNA用于特异性扩增含有pIX的RNA的5’端,所述引物是pIXrev和嵌套引物pIXrev-N2。对扩增的片段进行克隆和测序提供了转录起始位点的位置及含有pIX序列的mRNA的5’序列。在这种方式中,鉴别了可能的pIX编码mRNA。pIX表达水平与相应的重组腺病毒的稳定性之间的关系因此也可以确定。
实施例11:来自B群腺病毒的E1B和pIX序列
上述实施例使用Ad35病毒作为实例。彼此相当同源的B亚群的其它成员可能具有相似的pIX调节。
为对此进行研究,我们将B亚群成员的E1B和pIX序列进行序列对比。Ad7的序列(SEQ ID NO:57)通过Genbank登记号X03000获得。Ad11的序列(SEQ ID NO:56)通过与针对Ad35序列(SEQ ID NO:55)所述(WO00/70071)相似的Lark技术(UK),对分离自Ad11p野生型病毒的DNA序列进行鸟枪法(shotgun)测序而揭示。Ad11和Ad35是彼此高度同源的(整体98.1%相似性),主要差别位于六邻体和尾丝结(knob)中。Ad11序列也在WO02/053759中揭示。E1A的聚腺苷酸化位点(pA)与pIX基因的pA下游之间的序列用于序列对比(图10)。Ad35与Ad11的整体相似性(在这个区域中)为98.4%,与Ad7的整体相似性为82.9%。这使得在这些病毒中pIX的表达是以相同方式调节是非常可能的。
因此,在先前的实施例中例证的本发明的方法和手段可以用于增加亚群B的其它重组腺病毒的稳定性和/或插入容量,在此通过Ad11和Ad7例证。
实施例12:在Ad5互补细胞系上表达Ad5-E4/Orf6的缺失E1的Ad35病毒的产生
腺病毒血清型35基因组的测序以及基于质粒的载体系统的构建和重组的基于Ad35的病毒的产生已经在WO00/70071中详细描述。
如下将Ad5-E4-orf6编码序列克隆进pAdApt35IP1(ECACC保藏号P02041228,关于这个质粒的克隆详见于WO00/70071)中。将该质粒用NheI和AvrII消化并用牛小肠磷酸酶(New England Biolabs)去磷酸化。消化的DNA使用GeneClean试剂盒从凝胶中分离。将质粒pΔMT.Orf6.Hygro(图11,ECACC保藏号P02041226)用NheI消化,随后用XbaI部分消化。在凝胶上分离所得条带后,将与E4-orf6序列连接的ΔMT启动子相应的1350bp片段从凝胶中纯化。接着,将这两个分离的片段连接并转化进电感受态DH10B细胞(Invitrogen/LifeTechnologies),之后,选择具有与SV40poly(A)信号方向正确的插入体的一个集落进行大规模DNA制备。这样产生了构建体pAd35.ΔMT.Orf6(图12),其含有与突变的金属硫蛋白(ΔMT)启动子功能性连接的Ad5 E4-orf6编码序列。ΔMT启动子已经由Hagmeyer et al.(1996)描述。Ad5 E4-orf6序列相应于Ad5序列中的第33193-34077位核苷酸(Genbank登记号M73260)。为测试Ad5 E4-orf6蛋白的表达是否使得完全缺失E1的Ad35载体可能在Ad5互补细胞上生产,将pAd35.ΔMT.Orf6与Ad35主链构建体pWE.Ad35.pIX-rITR在PER.C6细胞上共转染。为此,将pAd35.ΔMT.Orf6用PI-Psp-1消化并将pWE.Ad35.pIX-rITR用NotI消化以从主链中释放腺病毒插入体。将2μg消化的pAd35.ΔMT.Orf6和6μg消化的pWE.Ad35.pIX-rITR使用LipofectAmine转染。将该转染混合物加入在前一天以3.5×106个细胞/T25培养瓶的密度接种的PER.C6中。第二天,将培养基更换为PER.C6培养基(具有10%FBS和10mMMgCl2的DMEM)并将细胞在37℃/10%CO2中进一步温育。对照转染使用pAdApt35.Luc与pWE.Ad35.pIX-rITR共转染及单独的pWE.Ad35.pIX-rITR进行。在转染两天后,将细胞从T25传递至T80培养瓶并入上述温育。再三天后,用pAd35.ΔMT.Orf6与Ad35主链一起转染的培养物示出表示病毒复制的致细胞病变作用(CPE),在进一步温育2天后收获(包括细胞和培养基)。将细胞悬浮液进行2次冷冻/解冻循环,并将所得材料(粗制裂解物)在-20℃保存直至进一步使用。其它培养瓶未示出CPE,在移至T80中6天后在T80培养瓶中以1∶3传代。再过5天后,pAdApt35.Luc+pWE.Ad35.pIX-rITR转染的培养瓶示出少量的类CPE事件,但不进一步进展。将得自pAd35.ΔMT.Orf6转染的0.2和0.5ml的粗制裂解物用于再次感染于T80培养瓶中大约85%铺满的PER.C6细胞。这样在温育一天后产生完全CPE,表明所述粗制裂解物中存在感染性病毒。这些培养物也通过两次冷冻/解冻循环收获。单独用构建体pAd35.ΔMT.Orf6在PER.C6细胞上进行另外的对照转染,以证实orf6自身的表达不产生细胞毒性及类CPE细胞死亡。总之,只有用pAd35.ΔMT.Orf6与pWE.Ad35.pIX-rITR一起转染才产生CPE及病毒复制。
进行PCR分析以证实用Ad5-E4-orf6置换从前的E1区的基于Ad35的病毒基因组的存在。为此,将病毒DNA如下从粗制裂解物中分离。将275μl的粗制裂解物与10μlDNaseI(10mg/ml)在37℃温育30分钟。随后,加入6.0μl 0.5MEDTA(pH8.0)、7.5μl 20%SDS和1.5μl20mg/ml蛋白酶K并涡旋混合。然后将该混合物在50℃温育1小时。最后,使用GeneClean Spin试剂盒(Bio 101,Inc.)分离病毒DNA。将2μl分离的DNA使用引物35psi-For和35R4进行PCR扩增。程序设定为94℃2分钟,随后进行30次(94℃30秒,58℃30秒及72℃5分钟)循环,最后在72℃10分钟结束反应。所述引物特异于Ad35序列并产生一个2.9kb的片段,范围从包装序列至第4669位核苷酸(编号如野生型Ad35序列),其因此包括Ad5 orf6转基因盒。对获得的PCR片段进行电泳示出该片段具有期望的长度,与在衔接质粒pAd35.ΔMT.Orf6上产生的对照PCR片段匹配。因此,如果病毒也表达Ad5-E4orf6,则可以在Ad5互补细胞上产生完全缺失E1的基于Ad35的载体。
实施例13:pWE.Ad35.pIX-rITR5E4的构建
将第一个PCR片段使用引物DF35-1和35FR扩增。扩增使用pWE.Ad35.pIX-rITR(见WO00/70071)作为模板DNA,使用Pwo DNA聚合酶(Roche)及另外的DMSO(Sigma,终浓度3%)进行。程序如下:94℃2分钟,随后进行30次(94℃30秒,52℃30秒及72℃3分钟)循环,最后的步骤是72℃8分钟以保证完整的片段。扩增产生相应于Ad35序列的第30224-31805位核苷酸的1.6kb的片段。将BamHI位点导入3’端。将扩增的DNA使用GeneClean试剂盒从凝胶中纯化并与pCRScript/Amp克隆载体试剂盒(Stratagene)连接。在转化入电感受态DH10B细胞中后,选择白色集落进行进一步分析。这样产生构建体pCR-fiber35。由于平端克隆,该PCR片段可以2个方向插入。选择在5’端pCRScript/Amp载体多接头中具有BamHI位点的插入体的克隆。用BamHI消化因此产生一个1.6kb的片段。测序证实PCR片段的正确扩增。将第二个PCR片段使用引物5E4F和5E4R扩增。用pWE.Ad5.AflII-rITRsp进行扩增,其是含有pWE.Ad5.AflII-rITR(ECACC保藏号P97082116,WO02/40665所述)中的一个额外的PacI位点的一种粘粒载体。pWE.Ad5.AflII-rITRsp作为模板,使用上述PwoDNA聚合酶,尽管pWE.Ad5.AflII-rITR也可以用于相同目的。在从凝胶中纯化后,将该DNA用SstI和BamHI(在PCR期间导入的两个位点)消化并将3kb的片段使用GeneClean试剂盒从琼脂糖凝胶中纯化。扩增的Ad5E4区域相应于Ad5序列的第32794至35828碱基对。第三个PCR片段使用引物355ITR和353ITR在pWE.Ad35.pIX-rITR上产生。如上所述进行PCR扩增。SstI位点(5’端)和EcoRI位点(3’端)位于所得160bp片段两侧。在如上述从凝胶中纯化后,将DNA用SstI和EcoRI消化。然后将相应于Ad35的右侧ITR的160bp的片段在低熔点的琼脂糖凝胶上从消化的末端中分离并在凝胶中收集。接着,将pUC119用BamHI和EcoRI消化并将3.1kb的片段使用GeneClean试剂盒从凝胶中分离。然后将上述处理的第二个和第三个PCR片段与BamHI/EcoRI消化的pUC119连接,产生pUC.Ad5E4-35ITR。对克隆的PCR衍生的插入体进行测序以核实正确的扩增。接着,将pCR-fiber35中的1.6kb插入体用BamHI切除并将该片段如上述从凝胶中纯化。将pUC.Ad5E4-35ITR也用BamHI消化并将该线性片段从凝胶中纯化。将这两个片段连接并选择具有相互正确方向的克隆,产生pUC.35-5E4(图14)。pUC.35-5E4的构建步骤示于图13。将pUC.35-5E4中的腺病毒插入体亚克隆进pBr.Ad35.PRn中(图15;见WO00/70071),这是具有Ad353’序列的一种构建体。为此,将构建体pUC.35-5E4用MluI和NotI消化并使用GeneClean试剂盒将4.7kb片段从凝胶中纯化。然后将这个片段与得自经MluI和NotI消化的构建体pBr.Ad35.PRn的载体片段连接。将这个16.3kb的片段使用琼脂糖酶(Roche)从凝胶中纯化。然后将该连接物转化进感受态DH10B细胞中。所得构建体称为pBr.Ad35.PR5E4(图16,ECACC保藏号P02041229)。最后的步骤是需将修饰的Ad35序列的3’末端克隆进病毒粘粒克隆pWE.Ad35.pIX-rITR中。为此,将两个片段在λ噬菌体包装反应(Stratagene)中根据厂商说明书组合。第一个片段是通过用PacI和SwaI消化的得自pBr.Ad35.PR5E4的16.8kb的修饰的Ad35插入体,第二个片段是通过用PacI和SwaI消化pWE.Ad35.pIX-rITR获得的22.8kb的片段。这两个片段的正确组合产生pWE.Ad35.pIX-rITR5E4(图17)。因此在这个构建体中,Ad35主链中的E4区由衍生自Ad5的相应区域置换。
实施例14:pWE.Ad35.pIX-rITR5Orf6的构建
为获得含有从pIX基因(Ad35序列中的第3401位核苷酸)至右侧ITR的末端的Ad35序列,而且E4-orf6和-orf6/7的序列交换为Ad5的相应序列的一种腺病毒主链构建体,将Ad35和Ad5序列经PCR扩增并如下述组合。PCR片段使用添加直至3%的DMSO的Pwo DNA聚合酶产生。第一个PCR使用pBr.Ad35.PRn(图15;见WO00/70071)作为模板及使用引物E4-F1和E4-R2。其程序设定为:94℃2分钟,随后进行5次(94℃30秒,50℃30秒及72℃1分钟)循环,接着进行30次(94℃30秒,60℃30秒及72℃1分钟)循环,最后在68℃8分钟结束反应。将所得1.8kb的片段使用GeneClean试剂盒纯化。第二个PCR使用pWE.Ad5.AflII-rITRsp作为模板,其是含有pWE.Ad5.AflII-rITR(ECACC保藏号P97082116,见WO02/40665所述)中一个PacI位点的粘粒载体,并使用引物E4-F3和E4-R4进行。其程序设定如下:94℃2分钟,随后进行30次(94℃30秒,62℃30秒及72℃1分钟)循环,最后在68℃8分钟结束。将1.1kb片段如上纯化。第三个PCR用pBr.Ad35.PRn作为模板及使用引物E4-F5和E4-R6进行。其程序设定如下:94℃2分钟,随后进行5次(94℃30秒,48℃30秒及72℃45秒)循环,接着进行30次(94℃30秒,56℃30秒及72℃45秒)循环,最后在68℃8分钟结束。将366bp片段如上述纯化。将纯化的片段的样品加样于凝胶上以估计浓度,然后将该片段混合在一起,含有700ng PCR-1、650ng PCR-2和430ng PCR-3,共30μl。向此混合物中加入3μlEcoPol缓冲液(New England Biolabs),3μl 2mM dNTP溶液及3μl milliQ H2O。将所得混合物在94℃温育3分钟,然后在一个PCR设备中以0.5℃/秒的速度冷却至65℃。在65℃温育10分钟后,将该混合物以0.05℃/秒的速度进一步冷却至20℃并在20℃温育10分钟。然后加入1μl(5单位)Klenow酶(NewEngland Biolabs),随后在37℃温育60分钟。将5μl的这种Klenow混合物用作模板以如下单独扩增两个片段。在一个反应中使用引物组1:NF-1和NcoI-R,使用Pwo DNA聚合酶(Roche),加入DMSO至终浓度为3%并使用如下PCR程序:94℃2分钟,随后进行30次(94℃30秒,66℃30秒及72℃3分钟)循环,最后在68℃8分钟结束。在一个反应中使用引物组2:NcoI-F和NR-2,使用Pwo DNA聚合酶(Roche),加入DMSO至终浓度为3%并使用如下PCR程序:94℃2分钟,随后进行30次(94℃30秒,62℃30秒及72℃90秒)循环,最后在68℃8分钟结束。将所得2.7kb片段(引物组1)口1.1kb片段(引物组2)使用GeneClean试剂盒从凝胶中纯化,并均与pCRscriptAmp载体(Stratagene)连接及转化进DH10B电感受态细胞中。这样产生构建体pCRscriptAmp.NFI-NcoIR(图18)和构建体pCRscriptAmp.NcoIF-NR2(图19)。由于插入体含有平端,因此每个克隆获得两个方向。使用KpnI消化,选择具有进一步克隆所需方向的构建体(见图18和19)。然后对插入体进行测序以核实正确的扩增。接着,将来自pCRscriptAmp-NcoIF-NR2的部分插入体使用BamHI和NcoI切除,并如上述从凝胶中纯化。将pCRscriptAmp-NFI-NcoIR用相同的酶消化并将含有载体的片段也从凝胶中纯化。将这些片段连接产生pCR.NF1-NR2(图20)。pCR.NF1-NR2含有Ad35序列的第30162-33234位核苷酸之间的Ad35序列及第31879-32974位核苷酸之间的E4-orf6和E4-orf6/7序列置换为位于Genbank中公布的Ad5序列(登记号M73260)第32968至34077位之间的Ad5衍生序列。因此,正如从图21和22所示氨基酸序列对比中可见,克隆的E4-orf6蛋白的氨基酸序列与在Ad5(SEQ ID NO:61;Ad35的E4-orf6的氨基酸序列为SEQ ID NO:62)中发现的E4-orf6序列相同,而且E4-orf6/7氨基酸序列与Ad5中存在的E4-orf6/7序列绝大部分相同(Ad 5的E4-orf6/7序列为SEQ ID NO:63,Ad35的E4-orf6/7序列为SEQ IDNO:64,克隆的融合蛋白为SEQ.ID.NO.65)。显然,使用不偏离本发明的上述一般方法可以设计不同的杂合Ad35-Ad5 E4构建体。然后将来自pCR.NF1-NR2的这种嵌合插入体克隆进pWE.Ad35.pIX-rITR:将pCR.NF1-NR-2用MluI和NdeI消化并将所得2.8kb片段使用GeneClean试剂盒从凝胶中纯化。将构建体pBr.Ad35.PRn也用MluI和NdeI消化并将18kb载体片段使用琼脂糖酶(Roche)从凝胶中分离。将这两个片段连接产生构建体pBr.Ad35.PR.5Orf6(图23,ECACC保藏号P02041227)。将这个构建体中含有嵌合E4区域的PacI和SwaI之间的Ad35序列使用上述λ噬菌体包装克隆进构建体pWE.Ad35.pIX-rITR中。所得pWE.Ad35pIX-rITR.5Orf6(图24)通过在PER.C6包装细胞上与Ad35衔接质粒共转染用于产生重组的基于Ad35的病毒。
实施例15:构建pWE.Ad35.pIX-rITRΔE3.5E4和pWE.Ad35.pIX-rITRΔE3.5Orf6
将Ad35主链通过缺失E3序列而进一步修饰。已知E3蛋白调节宿主对腺病毒感染的免疫应答并因此对重组病毒的体外增殖是非必需的。另外,E3序列的缺失使得可以在载体中插入一个更大的异源序列而不损害包装效力。同样,就腺病毒载体用作免疫接种的载体而言,由E3区编码的免疫调节基因不是优选的。
如前述构建pWE.Ad35.pIX-rITRΔE3(图1)。为构建上述E3缺失形式的E4修饰的主链构建体,如下在pBr.Ad35.PRnΔE3(图25)构建体中导入E4修饰。将构建体pUC.35-5E4(图13)用MluI和NotI消化并将4.7kb片段使用GeneCleanII试剂盒从凝胶中分离。将构建体pBr.Ad35.PRnΔE3也用MluI和NotI消化并将13.6kb载体片段使用GeneClean spin试剂盒从凝胶中分离。将这些片段连接产生构建体pBr.Ad35.ΔE3.PR5E4(图26)。将构建体pCR.NF1-NR2(图20)用MluI、NdeI和BglI消化(后者将载体片段消化为较小片段),并将2.8kb片段使用GeneClean spin试剂盒从凝胶中分离。将构建体pBr.Ad35.PRnΔE3用MluI和NdeI消化,使用CIP酶(New EnglandBiolabs)去磷酸化并将15.2kb载体片段使用GeneClean spin试剂盒从凝胶中分离。将这些片段连接产生构建体pBr.Ad35.ΔE3.PR5Orf6(图27)。
然后将pBr.Ad35.ΔE3.PR5E4和pBr.Ad35.ΔE3.PR5Orf6用于将pWE.Ad35.pIX-rITR中的3’PacI-SwaI片段交换为pBr.Ad35.ΔE3.PR5E4和pBr.Ad35.ΔE3.PR5Orf6中的相应区域。这样产生构建体pWE.Ad35.pIX-rITRΔE3.5E4和pWE.Ad35.pIX-rITRΔE3.5Orf6。产生这些大粘粒的另一种方法是在连接反应中使用三个片段包装:一个来自pWE.Ad35.pIX-rITR的14.7kb的NotI-PacI片段、来自pBr.Ad35.ΔE3.PR5E4或pBr.Ad35.ΔE3.PR5Orf6的PacI-NotI插入体及NotI消化的pWE15粘粒载体片段(Stratagene)。后一片段也可以分离自NotI/PacI消化的pWE.Ad35.pIX-rITR。例如NotI-消化的pWE.Ad35.pIX-rITRΔE3.5Orf6与例如PI-PspI消化的pAdApt35.LacZ.rsv(实施例9)共转染进PER.C6TM细胞中将产生衍生自Ad35的重组腺病毒,该重组腺病毒包含衍生自Ad5的E4-orf6(授予在PER.C6上增殖的能力),所述重组腺病毒进一步具有一个异源pIX启动子,导致pIX表达水平升高及产生稳定的病毒体。
实施例16:在PER.C6细胞上产生E1和E1/E3缺失的基于Ad35的载体
为使用基于pBr.Ad35.PRn的构建体能在互补细胞系PER.C6上产生重组Ad35病毒,我们首先产生含有Ad35序列(WO00/70071)的第3401至24650碱基对的Ad35序列的一种新的构建体,并因此与衔接质粒和基于pBr.Ad35.PRn的构建体重叠。将这三个质粒转染进PER.C6细胞中,进行双同源重组产生一个完整的病毒基因组,重组病毒的复制如图18所示。需要的质粒通过缺失pWE.Ad35.pIX-rITR中的大部分Ad35序列而产生。为此,将pWE.Ad35.pIX-rITR用EcoRV消化并将含有29kb载体的片段使用Geneclean spin试剂盒从低熔点凝胶中纯化。将纯化的DNA自身连接并用于转化DH10B电感受态细菌(Invitrogen/LTI)产生pWE.Ad35.pIX-EcoRV(图29)。
用于转染的所有DNA均如表V所示消化,在65℃热失活15分钟,用于转染中时不用进一步处理。将PER.C6细胞在转染前一天以3×106个细胞/培养瓶的密度接种于T25培养瓶中并如表V所示使用LipofectAmine(Invitrogen/LTI)根据厂商说明书进行转染,除了在5小时后将无血清DMEM培养基(Gibco/BRL)中转染混合物用PER.C6培养基(DMEM,10%FBS和10mM MgCl2)置换。第2天,通过荧光显微镜估计转染效力为50%。两天后,将细胞经胰蛋白酶消化并再接种于T80培养瓶中并在37℃/10%CO2中进一步温育。在转染6天后,所有培养物均示出完全致细胞病变作用(CPE,表示病毒增殖),除了用Ad35.AdApt.eGFP+pWE.Ad35.pIX-rITR转染的PER.C6培养物。一天后,收获具有CPE的培养瓶中的细胞和培养基并进行两次冷冻/解冻循环,通过离心(10分钟,1500rpm)从细胞碎片中澄清,并将100μl这些粗制裂解物用于再次感染于T80培养瓶中85%铺满的新鲜PER.C6细胞。通过胰蛋白酶消化收获没有示出CPE迹象的Ad35.AdApt.eGFP+pWE.Ad35.pIX-rITR转染物。感染新鲜PER.C6细胞2天后,所有培养瓶均示出了完全CPE,除了一个在开始收获时没有示出CPE迹象。这清楚示出当Ad5 E4-orf6基因产物从Ad35主链中表达时,完全缺失E1的基于Ad35的病毒可以在PER.C6细胞上生产。
实施例17:具有驱动pIX表达的异源启动子的缺失E1的Ad35病毒
为研究在完全缺失E1的病毒中异源启动子序列激活pIX基因的作用,使用一系列衔接质粒产生重组Ad35病毒。为此,将pAdApt35LacZ、pAdApt35.LacZ.rsv(实施例9)、pAdApt535.LacZ(实施例5)和pAdApt35BLacZ(在pIX基因的前面含有Ad35E1B启动子序列;见下述)用pIPsp-1消化并如实施例2(及WO00/70071)所述用于产生具有NotI消化的粘粒pWE/Ad35-3481和pWE/Ad35-3481ΔE3(实施例7)的病毒。另外,产生具有衔接质粒pBr.Ad35.ΔSM.AdAptLacZ(图7;实施例10)的病毒。这个衔接质粒缺失E1A和大部分E1B序列。其保留0.6kb的3’E1B-55K序列及还具有55K的终止密码子与pIX的起始密码子之间的wt序列。
在完全CPE时,收获细胞和培养基,冷冻/解冻并离心以除去细胞碎片。然后将每个转染物的上清(澄清的裂解物)用于进行如实施例9所述的噬斑分析。将澄清的裂解物系列稀释10倍并将10-5至10-9的稀释液铺板。
在加入琼脂覆层后一周,噬斑变得可见并将其用X-gal染色以监测LacZ活性。表VI概括示出了这些实验的结果。具有除了调节pIX的内源近端pIX上游序列的额外或其它序列所有Ad35病毒进行的更好,在我们的分析中与E1缺失的Ad35.AdApt.LacZ病毒相比具有较高数目的表达噬斑。注意Ad35.ΔSM.LacZ病毒的基因组总长度(是野生型Ad35的106%)超过针对Ad5病毒(105%)测定的最大可包装长度。这也许影响针对这个病毒获得的结果。Ad35.AdApt.LacZ.rsv(105%)也在理论上可包装大小的边界。综合这些结果示出驱动pIX表达的一个异源启动子改善最大耐受的包装大小及E1缺失的Ad35病毒的稳定性。针对具有较长内源近端序列的病毒(Ad35.ΔSM.LacZ)也同样,这提示在此额外的(E1B 55K)序列含有pIX表达的调节序列。
pAdapt35BLacZ是具有调节pIX基因的Ad35E1B启动子序列的一个Ad35衔接质粒。
衔接质粒pAdApt35BLacZ如下产生。将E1B启动子片段使用引物35E1Blong和Ad35E1Bpromrev扩增。这两个引物均被磷酸化。使用Pwo DNA聚合酶(Inno-train,Diagnostic GmbH)根据厂商说明书进行反应。pBr.Ad35.leftITR-pIX用作模板DNA(25ng,见WO02/40665所述)。该程序设定如下:94℃2分钟,然后进行30次(94℃30秒,60℃30秒及72℃1分钟)循环,最后在72℃10分钟结束。冷却/加热坡度设定为2℃/秒。这个PCR导致125个核苷酸的Ad35的潜在的E1B启动子扩增。然后将构建体pAdapt535.LacZ(实施例5)用MfeI和BglII消化。在消化后,将该载体用Klenow酶处理以产生平端。去磷酸化步骤使用SAP(Roche)进行。然后将如此处理的8kb载体片段从凝胶中分离。将E1B启动子区域也从凝胶中分离。连接这两个片段并转化进DH5α-Tlr感受态细胞中(Invitrogen)。所得质粒中E1B启动子的正确方向通过用HpaI和ApaLI消化而证实。在选择正确的克隆后,插入的E1B启动子序列也通过测序加以核实。
实施例18:pIX基因的启动子位于Ad35和Ad11病毒中E1B-55K编码序列的3’端。
基于上述结果,我们期望亚群B病毒中的pIX启动子位于E1B-55K编码区。为对此加以研究,我们如实施例10所述开始鉴别pIX mRNA加帽位点。为此,使用wtAd35、wtAd11和Ad35.E1B+.AdApt.Luc病毒以50VP/细胞的MOI感染PER55K克隆16细胞。取wtAd5作为对照,因为这个病毒的启动子和mRNA起始位点已知。在感染后16-18小时,使用TRIzol试剂(Invitrogen)根据厂商说明从感染的培养物中分离RNA。在该程序结束时,将分离的RNA贮存在100%甲酰胺中。使用GeneRacer试剂盒(Invitrogen)扩增pIX转录物的5’末端,以确定转录起始的位置。在开始GeneRacer方案之前,如GeneRacer方案所述将5μgRNA通过乙酸钠沉淀从甲酰胺中纯化。将纯化的RNA根据厂商方案进行处理以扩增pIXmRNA的5’末端。在磷酸酶处理和随后用烟草酸焦磷酸酶除去帽结构及与Generacer RNA oligo连接后,使用来自该试剂盒的SuperScriptTMII反转录酶合成cDNA。CDNA通过使用pIX的一个基因特异性(反向)引物的反转录合成。就Ad35(wt及E1B+.Luc病毒)和wtAd11而言,使用引物pIXrev,就Ad5而言,使用引物pIXrev-Ad5。所得cDNA(1μl,未知浓度)用作PCR模板以产生dsDNA。这个PCR使用Pwo DNA聚合酶(Roche)根据厂商说明书进行,并添加DMSO(Sigma;3%v/v)。所述扩增使用来自该试剂盒的特异于与所述mRNA的5’端连接的寡核苷酸的GeneRacer5’引物,及如上述基因特异性反向引物进行。反应条件如下:在94℃变性2分钟,随后进行30次(94℃30秒,60℃30秒,72℃2分钟)循环,最后在68℃延长8分钟。所得DNA片段通过在1.0%琼脂糖凝胶上电泳而按大小分离。就Ad5而言,获得一个480bp的片段,就Ad35(两个病毒)和Ad11而言,获得一个200bp的片段。Ad11还示出一个2kb片段。切下所有片段并从琼脂糖凝胶中纯化。将纯化的DNA片段克隆进pCR4Blunt-TOPO载体(Invitrogen)中。对此载体使用商业M13正向和反向引物进行测序。将所得序列与野生型序列相对比以确定pIX转录起始的位置。从Ad35和Ad11 cDNA制品中分离的200bp条带组成真正的pIXmRNA,分离自Ad11的2kb片段源于E1B启动子。图30示出Ad35(SEQ ID NO:59)和Ad11(SEQ ID NO:58)cDNA序列与wt Ad35序列(SEQ.ID.NO.60)的对比。该对比揭示了pIX mRNA中剪接的内含子序列的位置。图31示出Ad35的E1B-pIX区域中鉴别的加帽位点和剪接位点的位置。就Ad5而言,鉴别了期望的pIX mRNA(Babiss andVales,1991)的加帽位点位于第3580位(未示出,编号如Genbank登记号M73260)。就Ad35而言,所述加帽位点位于E1B-55K基因3’末端第3339位的A残基上(Ad35序列,WO00/70071)。在Ad11中,所述加帽位点相似地在T残基上发现(Genbank登记号AY163756第3339位)。令人感兴趣地,在pIX的启动子位于在Ad5病毒中之处,55K基因的终止密码子和pIX的起始密码子之间的序列在Ad35和Ad11病毒中被从mRNA中剪接掉。这些结果提供了有利证据表明在Ad35和Ad11中,pIX基因表达是由位于55K基因的3’末端的一个启动子调节的。
实施例19:保留3’E1B-55K序列的一段短序列的E1缺失的Ad35病毒具有较大的包装能力
随着pIX mRNA加帽位点的鉴别,在病毒载体中包含天然的Ad35启动子以正确表达pIX以及尽可能多地限制E1B-55K序列成为可能。在此作为一个非限制性实例,我们揭示了保留166bp的55K编码序列的3’末端的Ad35衔接质粒的构建(pAdApt35Bsu.Luc)及稳定性和/或包装能力增加的E1缺失的Ad35Luc病毒的产生。这个166bp的序列不编码一个功能性55K基因产物,但在其相对于pIX编码序列的天然位置中含有在前述实施例中鉴别的pIX mRNA加帽位点。
如下产生构建体pAdApt35Bsu.Luc:
首先使用40ng pBr.Ad35.leftITR-pIX作为靶DNA(见WO02/40665所述)及使用引物Bsu55KF和Age-pIXR产生一个PCR片段。该PCR使用Pwo DNA聚合酶(Genaxis)根据厂商说明书进行。另外,使用3%v/v DMSO(Sigma)。程序设定如下:94℃2分钟,然后进行30次(94℃30秒,60℃30秒及72℃1.5分钟)循环,最后在68℃8分钟结束。将所得1.2kb产物根据厂商的方案直接克隆进pCR4Blunt-TOPO载体(Invitrogen)中,产生pCR4Blunt-TOPO.Bsu-Age。将该构建体通过用PvuII(New EnglandBiolabs)消化进行检测。将Bsu-Age片段从用Bsu36I(New EnglandBiolabs)消化及用Klenow酶(New England Biolabs)处理以产生平端的pCR4Blunt-TOPO.Bsu-Age质粒中分离。然后将该DNA使用PCR纯化试剂盒(Qiagen)纯化及用AgeI(New England Biolabs)消化。将1kb的片段使用Gene clean II试剂盒(BiolOl,Inc.)从凝胶中分离。平行地,将构建体pAdApt35.Luc(见WO00/70071所述)用BglII消化并用Klenow酶处理。将该DNA使用PCR纯化试剂盒(Qiagen)纯化。纯化的DNA用AgeI(New England Biolabs)消化及用SAP(Roche)去磷酸化。将5.8-Kb片段使用Gene clean II试剂盒(Bio101)从凝胶中分离。将这两个片段在连接反应中以等摩尔量混合并转化进T1抗性EMDH10B细胞(Invitrogen)中。这样产生了质粒pAdApt35Bsu.Luc。
为产生E1-缺失的病毒,将pAdApt35Bsu.Luc用pIPsp-I消化并与NotI消化的pWE.Ad35-3481(实施例7)及前述的pWE.Ad35-3481ΔE3在PER55K克隆16细胞上共转化。pWE.Ad35-3481ΔE3含有与pWE.Ad35.pIX-rITRΔE3(实施例2)相同的E3缺失,根据实施例7所述方法使用构建体pWE.Ad35-3481ΔNdeI和一个来自pWE.Ad35.pIX-rITRΔE3的26.6kb的NdeI片段产生。
另外,产生E1-缺失的Ad35病毒,其在病毒主链中含有来自Ad5的E4-Orf6和E4-Orf6/7序列代替天然的Ad35序列(见实施例16在未修饰的PER.C6细胞上产生这种病毒)。在本实施例中,将用pIPsp-I消化的pAdApt35Bsu.Luc与NotI/EcoRV消化的pWE.Ad35.pIX-EcoRV及与PacI/NotI消化的pBr.Ad35.PR5Orf6(有或无E3区)共转染。所有转染在转染后一周内均产生完全CPE,如上述收获细胞和培养基。然后将病毒进行噬斑纯化,并针对转基因区域的完整性通过PCR分析在合适的互补细胞上扩增的及源自单个噬斑的病毒原种(viral stocks)。如实施例3所述使用引物AdApt35CMVF和pIXrevN2进行转基因PCR。图32示出了对源自Ad35Bsu.Luc及源自Ad35Bsu.Luc.5Orf6病毒的噬斑进行PCR的结果实例。
尽管在病毒主链中存在E3区或E4-Orf6序列,但所有测试的噬斑(每个病毒5-10个)均含有一个完整的转基因。一个例外出现在来自Ad35Bsu.Luc病毒的一个噬斑,其示出可能源自具有少量缺失的病毒的大约1.6kb的一个模糊条带(图32;泳道12)。在所有病毒样品中均出现的大约500bp的模糊条带是引物在病毒主链上的背景条带。具有荧光素酶表达盒的含有E3的Ad35病毒在噬斑纯化后被证实是稳定的,这个观测结果与先前用Ad35.AdApt.Luc病毒获得的结果相反,该病毒完全缺失E1而且不含有所述166bp的3’55K编码序列。使用标准pAdApt35.Luc质粒,我们不能产生含有E3区的噬斑纯化的病毒。因此随着在主链中掺入额外的55K序列,我们现在可以产生总长度超过34.6kb但无明显的不稳定性的病毒。这与wt Ad35病毒的长度密切匹配。如果使用E3-缺失的主链,外源序列的容量理论上超过5kb。显然,在不偏离本发明范围内,可以比在现有的实施例中掺入更多的E1B-55K序列和/或将3’55K序列与异源增强子序列组合。
表I:在三层培养瓶中的克隆#16细胞上E1-和E1/E3-缺失的Ad35病毒的产量。
| 病毒 |
Scale(T175<sup>III</sup>培养瓶) |
Total#of VirusDSP后病毒颗粒总数 |
VP/细胞 |
| Ad35_AdApt.eGFP |
4 |
7.5×10<sup>11</sup> |
2500 |
| Ad35.ΔE3.AdApt.empty |
8 |
2×10<sup>12</sup> |
3300 |
| Ad35.ΔE3.AdApt.LacZ |
8 |
3.8×10<sup>11</sup> |
600 |
| Ad35.ΔE3.AdApt.MV-F |
4 |
8.8×10<sup>11</sup> |
2900 |
| Ad35.ΔE3.AdApt.MV-H |
8 |
2.6×10<sup>12</sup> |
4250 |
表II:对A549使用第二代病毒的粗制裂解物的转基因(LacZ)活性测试。%蓝色细胞代表用于感染的每种病毒的量。
| 病毒 |
10μl |
1μl |
0.1μl |
| Ad35.AdApt.LacZ.rsv |
95 |
15 |
<1 |
| Ad35.ΔE3.AdApt.LacZ.rsv |
90 |
10 |
<1 |
| Ad35.AdApt.LacZ.C4 |
2 |
<0.1 |
0 |
| Ad35.ΔE3.AdApt.LacZ.C4 |
15 |
<1 |
<0.1 |
表III:对A549使用第二代病毒的粗制裂解物的转基因(荧光素酶)活性测试。活性表示为相对光单位(RLU)。
| 病毒 |
10μl |
1μl |
0.1μl |
0.01μl |
| Ad35.AdApt.Luc.rsv |
845453 |
27940 |
178 |
26 |
| Ad35.ΔE3.AdApt.Luc.rsv |
258269 |
2217 |
46 |
6 |
| Ad35.AdApt.Luc.C4 |
6130 |
175 |
18 |
33 |
| Ad35.ΔE3.AdApt.Luc.C4 |
814642 |
6278 |
147 |
23 |
| Ad35.ΔE1AΔ21K.Luc |
1514698 |
50196 |
503 |
57 |
表IV:引物序列。
| 名称 |
序列 |
SEQ.ID.NO. |
| 35FR |
5′-CGGGATCCACTTTATTTTAGTTGTCGTCTTC-3′ |
1 |
| 35R4 |
5′-CGGAATTCTTAATTAAGGGAAATGCAAATCTGTGAGG-3′ |
2 |
| 35psi-For |
5′-GTGGTATTTATGGCAGGGTG-3′ |
3 |
| DF35-1 |
5′-CACTCACCACCTCCAATTCC-3′ |
4 |
| 5E4F |
5′-CGGGATCCGTTTGTGTTATGTTTCAACGTG-3′ |
5 |
| 5E4R |
5′-GCTGGCGAGCTCGGCGGAGTAACTTGTATGTG-3′ |
5 |
| 355ITR |
5′-GATCCGGAGCTCACAACGTCATTTTCCCACG-3′ |
7 |
| 353ITR |
5′-AGGAATTCGCGGCCGCATTTAAATC-3′ |
8 |
| E4-F1 |
5′-AGAGGAACACATTCCCCC-3′ |
9 |
| E4-R2 |
5′-GGGGAGAAAGGACTGTGTATTCTGTCAAATCG-3′ |
10 |
| E4-F3 |
5′-TTTGACAGAATACACAGTCCTTTCTCCCCGGCTGG-3′ |
11 |
| E4-R4 |
5′-ACAAAATACGAGAATGACTACGTCCGGCGTTCC-3′ |
12 |
| E4-F5 |
5′-GGACGTAGTCATTCTCGTATTTTGTATAGC-3′ |
13 |
| E4-R6 |
5′-TCACCAACACAGTGGGGG-3′ |
14 |
| NF-1 |
5′-CCACAACCCCCACTACTCCC-3′ |
15 |
| NR-2 |
5′-CGTCTCTTCCCTCTCCTCTCC-3′ |
16 |
| NcoI-R |
5′-AGGATCATCCGCTGCTGCCC-3′ |
17 |
| NcoI-F |
5′-CATCAGGATAGGGCGGTGG-3′ |
18 |
| 35E3for |
5′-AATGACTAATGCAGGTGCGC-3′ |
19 |
| 35E3rev |
5′-CGACGCGTTGTAGTCGTTGAGCTTCTAG-3′ |
20 |
| AdApt35CMVF |
5′-GTAGGTGTCAGCCTAGGTGGTC-3′ |
21 |
| 35pIXR |
5′-TCATGTCAGCTGCAAGACAG-3′ |
22 |
| SV40for |
5′-CAATGTATCTTATCATGTCTAG-3′ |
23 |
| pIX5Rmfe |
5′-CTCTCTCAATTGCAGATACAAAACTACATAAGACC-3′ |
24 |
| pIX35Fmfe |
5′-CTCTCTCAATTGTCTGTCTTGCAGCTGACATG-3′ |
25 |
| AdApt35pIXrev |
5′-CAATCTGTCCATCTGAAAATCC-3′ |
26 |
| pIXcosF-2 |
5′-CTGCTGGACGTCGCGGCCGCGACATGAGTGGAAATGCTTC-3′ |
27 |
| Adapt35-3 |
5′-TGCAAATCTGTGAGGGGAAC-3′ |
28 |
| 35D21 |
5′-TTA GAT CCA TGG ATC CCG CAG ACT C-3′ |
29 |
| 35B3 |
5′-CCT CAG CCC CAT TTC CAG-3′ |
30 |
| 35F1 |
5′-CGGAATTCTTAATTAATCGACATCATCAATAATATACCTTATAG-3′ |
31 |
| 35R4 |
5′-CGGAATTCTTCTTAATTAAGGGAAATGCAAATCTGTGAGG-3′ |
32 |
| Ad3555KMfeF |
5′-AACCAAGCTTCAATTGTCTCTGAA-3′ |
33 |
| Ad35pIXNcoR |
5′-CCACCCATGGCAGCTGCAAGACAG-3′ |
34 |
| Ad35pIXrev |
5′-TCAGCTCCAAGACAGAAAAAAC-3′ |
35 |
| Epr-F |
5′-GTGTTTACTTAAGGTGACGTC-3′ |
36 |
| Epr-R |
5′-GAAAGCCAGCTCCTATGAGC-3′ |
37 |
| pIXrev |
5′-GGCGGGTTGAACGGGTCTTCCA-3′ |
38 |
| pIXrev-N2 |
5′-GATGGGAGACGCCCTGTCAGATAAGG-3′ |
39 |
| 35E1Blong |
5′-AAGGTGACGTCAATATTTGTGTG-3′ |
40 |
| Ad35E1bpromrev |
5′-ATGAAAGCCAGCTCCTATGAG-3′ |
41 |
| pIXrev-Ad5 |
5′-AGGGGAGGAAGCCTTCAGG-3′ |
42 |
| Bsu55KF |
5′-AGG TGG GCG TAG AGG AAT G-3′ |
43 |
| Age-pIXR |
5′-CAA GAC GGG ATC TTG GCG G-3′ |
44 |
表V:实施例所述用于在PER.C6细胞上产生E1缺失的基于Ad35的病毒的构建体列表。衔接构建体用PacI消化,pWE.Ad35.pIX-EcoRV用NotI和EcoRV消化,E4-修饰的基于pBR的构建体用PacI和NotI消化。
| No. |
构建体 |
CPE |
| 1 |
pAdApt35.eGFP |
pWE.Ad35.pIX-EcoRV |
pBr.Ad35.PR5E4 |
有 |
| 2 |
pAdApt35.eGFP |
pWE.Ad35.pIX-EcoRV |
pBr.Ad35.PR5Orf6 |
有 |
| 3 |
pAdApt35.eGFP |
pWE.Ad35.pIX-EcoRV |
pBr.Ad35.ΔE3PR5E4 |
有 |
| 4 |
pAdApt35.eGFP |
pWE.Ad35.pIX-EcoRV |
pBr.Ad35.AE3.PR5Orf6 |
有 |
| 5 |
pAdApt35.eGFP |
pWE.Ad35.pIX-rITRxNotI |
无 |
| 6 |
pAdApt5.eGFP |
pWE.Ad5.AflII-rrTRxPacI |
有 |
表VI:具有不同的驱动pIX表达的启动子序列的Ad35病毒的LacZ阳性噬斑的百分比。(NP=无可见噬斑)
|
|
蓝色噬斑% |
|
| 病毒名称 |
有E3区 |
无E3区 |
病毒长度(包括E3区) |
| Ad35.AdApt.LacZ |
0% |
50% |
36,1kb |
| Ad535.AdApt.LacZ |
NP |
100% |
36,1kb |
| Ad35.AdAptB.LacZ |
5% |
80% |
36,2kb |
| Ad35.AdApt.LacZ.rsv |
90% |
100% |
36,5kb |
| Ad35ΔSM.LacZ |
50% |
100% |
36,7kb |
参考文献
Abrahamsen K,Kong H-L,Mastrangeli A,Brough D,Lizonova A,Crystal R and Falck-Pedersen E.(1997)Construction of an adenovirustype 7a E1A-vector.J Virol 11:8946-8951。
Babiss,L.E.and Vales,L.D.(1991).Promoter of the adenoviralpolypeptide IX gene:Similarity to E1B and inactivation by substitution ofthe simian virus TATA element.J.Virol.65(2),p598-605。
Bett,A.J.,Prevec,L.and Graham,F.L.(1993).Packaging capacityand stability of human adenovirus type 5 vectors.J.Virol.67(10),p5911-5921。
Boshart,M.,Weber,F.,Jahn,G.,Dorsch-Hasler,K.,FleckensteinB.and Schaffner W.(1985).A very strong enhancer is located upstreamof an immediate early gene of human cytomegalovirus.Cell 41,521-530。
Caravokyri,C.and Leppard,K.N.(1995).Constitutive episomalexpression of polypeptide IX(pIX)in a 293-based cell line complementsthe deficiency of pIX mutant adenovirus type 5.J.Virol.69(11),p6627-6633。
Fallaux,F.J.,Kranenburg,0.,Cramer,S.J.,Houweling,A.,vanOrmondt,H.,Hoeben,R.C.and van der Eb,A.J.(1996).Characterization of 911:A new helper cell line for the titration andpropagation of early region 1-deleted adenoviral vectors.Hum.GeneTher.7,p215-222。
Fallaux,F.J.,Bout,A.,van der Velde,I.,van den Wollenberg,D.J.,Hehir,K.M.,Keegan,J.,Auger,C.,Cramer,S.J.,van Ormondt,H.,van der Eb,A.J.,Valerio,D.and Hoeben,R.C.(1998).New helpercells and matched early region 1-deleted adenovirus vectors preventgeneration of replication competent adenoviruses.Hum.Gene Ther.9,p1909-1917。
Farina,S.F.,Gao,G.P.,Xiang,Z.Q.,Rux,J.J.,Burnett,R.M.,Alvira,M.R.,Marsh,J.,Ertl,H.C.and Wilson,J.M.(2001).Replication-defective vector based on a chimpanzee adenovirus.J.Virol.75(23),p11603-11613。
Francki,R.I.B.,Fauquet,C.M.,Knudson,L.and Brown,F.(1991).Classification and nomenclature of viruses.Fifth report of theinternational committee on taxonomy of viruses.Arch.Virol.Suppl.2,p140-144。
Furcinitti,P.S.,vanOostrom,J.and Burnett R.M.(1989).Adenoviruspolypeptide IX revealed as capsid cement by difference images fromelectron microscopy and crystallography.EMBO J.8(12),p3563-3570。
Grable,M.and Hearing,P.(1990).Adenovirus type 5packagingdomain is composed of a repeated element that is functionally redundant.J.Virol.Vol.64,No.5,p2047-2056。
Graham,F.O.,Smiley,J.,Russell,W.andNairn,R.(1970.Characteristics of a human cell line transformed by DNA from humanadenovirns type 5.J.Gen.Virol.36,p59-72。
Gustin,K.E.and Imperiale,M.J.(1998).Encapsidation of viral DNArequires the adenovirus LI 52/55-kilodalton protein.J.Virol.Vol 72,No.10,p7860-7870。
Ghosh-Choudhury,G.,Haj-Ahmad,Y.and Graham,F.L.(1987).Protein IX,a minor component of the human adenovirus capsid,isessential for the packaging of full length genomes.EMBO J.6(6),p1733-1739。
Hagmeyer BM,Duyndam MC,Angel P,De Groom RP,VerlaanM,Elfferich P,Van der Eb AJ and Zantema A.(1996)Oncogene 12:1025-1032。
Hehir,K.M.,Armentano,D.,Cardoza,L.M.,Chouette,T.L.,Berthelette,P.B.,White,G.A.,Couture,L.A.,Everton,M.B.,Keegan,J.,Martin,J.M.,Pratt,D.A.,Smith,M.P.,Smith,A.E.and Wadsworth,S.C.(1996).Molecular characterization of replication-competent variantsof adenovirus vectors and genome modifications to prevent theiroccurrence.J.Virol.70(12),p8459-8467。
Herrman,C.H.and Mathews,M.B.(1989).The adenovirusE1B19-kilodalton protein stimulates gene expression by increasing DNAlevels.Mol.Cell.Biol.9,p5412-5423。
Horwitz,M.S.(2001).Adenovirus immunoregulatory genes andtheir cellular targets.Virology 279(1),p1-8。
Leppard,K.N.(1997).E4 gene function in adenovirus,adenovirusvector and adeno-associated virus infections.J.Gen.Virol.78,p2131-2138。
Leppard,K.N.(1998).Regulated RNA processing and RNAtransport during adenovirus infection.Seminars in Virology 8,p301-307。
Lutz,P.,Rosa-Calatrava,M.andKedinger,C.(1997).The productof the adenovirus intermediate gene IX is a transcriptional activator.J.Virol.71(7),p5102-5109。
Pilder,S.,Logan,J.and Shenk,T.(1984).Deletion of the geneencoding the adenovirus type 5 early region 1B 21,000-molecular-weightpolypeptide leads to degradation of viral and host cell DNA.J.Virol.52(2),p664-671。
Pilder,S.,Moore,M.,Logan,J.and Shenk,T.(1986).Theadenovirus E1B 55K transformbing polypeptide modulates transport orcytoplasmic stabilization of viral and host cell mRNAs.Mol.Cell.Biol.6,p470-476。
Rao,L.,Debbas,M.,Sabbatini,P.,Hockenbery,D.,Korsmeyer,S.and White,E.(1992).The adenovirus E1A proteins induce apoptosis,which is inhibited by the E1B 19-kDa and Bcl-2 proteins.Proc.Natl.Acad.Sci.USA Vol.89,pp7742-7746。
Reese,M.G.and Eeckman,F.H.(1995).Novel neural networkalgorithms for improved eukaryotic promoter site recognition.Talk andAbstract from The seventh international genome sequencing and analysisconference.Hyatt Regency,Hilton Head Island,South Carolina,Sept16-20,1995。(http://www.fruitfly.org/seq_tools/prormoter.html)。
Robert,J.-J.,Gauffeny,I.,Maccario,J.,Jullien,C.,Benoit,P.,Vigne,E.,Crouzet,J.,Perricaudet,M.and Yeh,P.(2001).DegeneratedpIX-IVa2 adenoviral vector sequences lowers reacquisition of the E1genes during virus amplification in 293 cells.Gene Ther.8,p1713-1720。
Russell,W.C.(2000).Update on adenoviruses and its vectors.J.Gen.Virol.81,pp2573-2604。
Sambrook,J.and Russell,D.Molecular Cloning:A laboratorymanual.Third Edition.Cold Spring Harbor Press(2001).ISBN0-87969-576-5。
Shabram,P.W.,Giroux,D.D.,Goudreau,A.M.,Gregory,R.J.,Horn,M.T.,Huyghe,B.G.,Liu,X.,Nunnally,M.H.,Sugarman,B.J.and Sutjipto,S.(1997)Analytical anion-exchange HPLC ofrecombinant type-5 adenoviral particles.Hum.Gene Ther.8(4):453-465。
Shenk,T.(1996).Adenoviridae:The viruses and their replication.InVirology,eds.Fields,B.N.,Knipe,D.M.and Howley,P.M.(Lippincott-Raven,NewYork),Vol.2,pp2111-2148。
Van der Vliet,P.C.(1995).Adenovirus DNA replication.In Themolecular repertoire of adenoviruses II,eds.Doerfler,W.and Bohm,P.(Springer-Verlag,Berlin).Current Topics in Microbiology andImmunology 199/II,pp1-30。
White,E.,Grodzicker,T.and Stillman,B.W.(1984).Mutations inthe gene encoding the adenovirus early region 1B 19,000molecular-weight tumot antigen cause degradation of chromosomal DNA.J.Virol.52(2),p410-419。
Yew,P.R.and Berk,A.J.(1992).Inhibition of p53 transactivationrequired for transformation by adenovirus early region 1B protein.Nature 357,pp82-85。
Zhang,W.,Low,J.A.,Christensen,J.B.and Imperiale,M.I.(2001).Role for the adenovirus IVa2 protein in packaging of viral DNA.J.Virol.Vol.75No.21,pp10446-10454。
序列表
<110>克鲁塞尔荷兰公司
<120>稳定的腺病毒载体及其增殖方法
<130>0076WO00ORD
<150>EP02102631.5
<151>2002-11-25
<150>PCT/NL02/00281
<151>2002-04-25
<150>PCT/NL02/00656
<151>2002-10-15
<160>65
<170>PatentIn version 3.2
<210>1
<211>31
<212>DNA
<213>Artificial
<220>
<223>primer 35FR
<400>1
cgggatccac tttattttag ttgtcgtctt c 31
<210>2
<211>37
<212>DNA
<213>Artificial
<220>
<223>primer 35R4
<400>2
cggaattctt aattaaggga aatgcaaatc tgtgagg 37
<210>3
<211>20
<212>DNA
<213>Artificial
<220>
<223>primer 35psi-For
<400>3
gtggtattta tggcagggtg 20
<210>4
<211>20
<212>DNA
<213>Artificial
<220>
<223>primer DF35-1
<400>4
cactcaccac ctccaattcc 20
<210>5
<211>30
<212>DNA
<213>Artificial
<220>
<223>primer 5E4F
<400>5
cgggatccgt ttgtgttatg tttcaacgtg 30
<210>6
<211>32
<212>DNA
<213>Artificial
<220>
<223>primer 5E4R
<400>6
gctggcgagc tcggcggagt aacttgtatg tg 32
<210>7
<211>31
<212>DNA
<213>Artificial
<220>
<223>primer 355ITR
<400>7
gatccggagc tcacaacgtc attttcccac g 31
<210>8
<211>25
<212>DNA
<213>Artificial
<220>
<223>primer 353ITR
<400>8
aggaattcgc ggccgcattt aaatc 25
<210>9
<211>18
<212>DNA
<213>Artificial
<220>
<223>primer E4-F1
<400>9
agaggaacac attccccc 18
<210>10
<211>32
<212>DNA
<213>Artificial
<220>
<223>primer E4-R2
<400>10
ggggagaaag gactgtgtat tctgtcaaat gg 32
<210>11
<211>35
<212>DNA
<213>Artificial
<220>
<223>primer E4-F3
<400>11
tttgacagaa tacacagtcc tttctccccg gctgg 35
<210>12
<211>33
<212>DNA
<213>Artificial
<220>
<223>primer E4-R4
<400>12
acaaaatacg agaatgacta cgtccggcgt tcc 33
<210>13
<211>30
<212>DNA
<213>Artificial
<220>
<223>primer E4-F5
<400>13
ggacgtagtc attctcgtat tttgtatagc 30
<210>14
<211>18
<212>DNA
<213>Artificial
<220>
<223>primer E4-R6
<400>14
tcaccaacac agtggggg 18
<210>15
<211>20
<212>DNA
<213>Artificial
<220>
<223>primer NF-1
<400>15
ccacaacccc cactactccc 20
<210>16
<211>21
<212>DNA
<213>Artificial
<220>
<223>primer NR-2
<400>16
cgtctcttcc ctctcctctc c 21
<210>17
<211>20
<212>DNA
<213>Artificial
<220>
<223>primer NcoI-R
<400>17
aggatcatcc gctgctgccc 20
<210>18
<211>19
<212>DNA
<213>Artificial
<220>
<223>primer NcoI-F
<400>18
catcaggata gggcggtgg 19
<210>19
<211>20
<212>DNA
<213>Artificial
<220>
<223>primer 35E3for
<400>19
aatgactaat gcaggtgcgc 20
<210>20
<211>28
<212>DNA
<213>Artificial
<220>
<223>primer 35E3rev
<400>20
cgacgcgttg tagtcgttga gcttctag 28
<210>21
<211>22
<212>DNA
<213>Artificial
<220>
<223>primer AdApt35CMVF
<400>21
gtaggtgtca gcctaggtgg tc 22
<210>22
<211>20
<212>DNA
<213>Artificial
<220>
<223>primer 35pIXR
<400>22
tcatgtcagc tgcaagacag 20
<210>23
<211>22
<212>DNA
<213>Artificial
<220>
<223>primer SV40for
<400>23
caatgtatct tatcatgtct ag 22
<210>24
<211>35
<212>DNA
<213>Artificial
<220>
<223>primer pIX5Rmfe
<400>24
ctctctcaat tgcagataca aaactacata agacc 35
<210>25
<211>32
<212>DNA
<213>Artificial
<220>
<223>primer pIX35Fmfe
<400>25
ctctctcaat tgtctgtctt gcagctgaca tg 32
<210>26
<211>22
<212>DNA
<213>Artificial
<220>
<223>primer AdApt35pIXr
<400>26
caatctgtcc atctgaaaat cc 22
<210>27
<211>40
<212>DNA
<213>Artificial
<220>
<223>primer pIXcosF-2
<400>27
ctgctggacg tcgcggccgc gacatgagtg gaaatgcttc 40
<210>28
<211>20
<212>DNA
<213>Artificial
<220>
<223>primer Adapt35-3
<400>28
tgcaaatctg tgaggggaac 20
<210>29
<211>25
<212>DNA
<213>Artificial
<220>
<223>primer 35D21
<400>29
ttagatccat ggatcccgca gactc 25
<210>30
<211>18
<212>DNA
<213>Artificial
<220>
<223>primer 35B3
<400>30
cctcagcccc atttccag 18
<210>31
<211>44
<212>DNA
<213>Artificial
<220>
<223>primer 35F1
<400>31
cggaattctt aattaatcga catcatcaat aatatacctt atag 44
<210>32
<211>40
<212>DNA
<213>Artificial
<220>
<223>primer 35R4
<400>32
cggaattctt cttaattaag ggaaatgcaa atctgtgagg 40
<210>33
<211>24
<212>DNA
<213>Artificial
<220>
<223>primer Ad3555KMfeF
<400>33
aaccaagctt caattgtctc tgaa 24
<210>34
<211>24
<212>DNA
<213>Artificial
<220>
<223>primer Ad35pIXNcoR
<400>34
ccacccatgg cagctgcaag acag 24
<210>35
<211>22
<212>DNA
<213>Artificial
<220>
<223>primer Ad35pIXrev
<400>35
tcagctgcaa gacagaaaaa ac 22
<210>36
<211>21
<212>DNA
<213>Artificial
<220>
<223>primer Epr-F
<400>36
gtgtttactt aaggtgacgt c 21
<210>37
<211>20
<212>DNA
<213>Artificial
<220>
<223>primer Epr-R
<400>37
gaaagccagc tcctatgagc 20
<210>38
<211>22
<212>DNA
<213>Artificial
<220>
<223>primer pIXrev
<400>38
ggcgggttga acgggtcttc ca 22
<210>39
<211>26
<212>DNA
<213>Artificial
<220>
<223>primer pIXrev-N2
<400>39
gatgggagac gccctgtcag ataagg 26
<210>40
<211>23
<212>DNA
<213>Artificial
<220>
<223>primer 35ElBlong
<400>40
aaggtgacgt caatatttgt gtg 23
<210>41
<211>21
<212>DNA
<213>Artificial
<220>
<223>primer Ad35E1bpromrev
<400>41
atgaaagcca gctcctatga g 21
<210>42
<211>19
<212>DNA
<213>Artificial
<220>
<223>primer pIXrev-Ad5
<400>42
aggggaggaa gccttcagg 19
<210>43
<211>19
<212>DNA
<213>Artificial
<220>
<223>primer Bsu55KF
<400>43
aggtgggcgt agaggaatg 19
<210>44
<211>19
<212>DNA
<213>Artificial
<220>
<223>primer Age-pIXR
<400>44
caagacggga tcttggcgg 19
<210>45
<211>102
<212>DNA
<213>Adenovirus type 2
<220>
<221>misc_feature
<223>Ad2 proximal pIX upstream sequence
<220>
<221>E1B 55K stopcodon
<222>(1)..(3)
<220>
<221>Spl site
<222>(18)..(24)
<220>
<221>TATA_signal
<222>(45)..(51)
<220>
<221>pIX startcodon
<222>(99)..(102)
<400>45
tgaggtactg aaatgtgtgg gcgtggctta agggtgggaa agaatatata aggtgggggt 60
ctcatgtagt tttgtatctg ttttgcagca gccgccgcca tg 102
<210>46
<211>105
<212>DNA
<213>Adenovirus type 5
<220>
<221>misc feature
<223>Ad5 proximal pIX upstream sequence
<220>
<221>E1B 55K stopcodon
<222>(1)..(3)
<220>
<221>Spl site
<222>(18)..(24)
<220>
<221>TATA_signal
<222>(45)..(51)
<220>
<221>pIX startcodon
<222>(103)..(105)
<400>46
tgaggtactg aaatgtgtgg gcgtggctta agggtgggaa agaatatata aggtgggggt 60
cttatgtagt tttgtatctg ttttgcagca gccgccgccg ccatg 105
<210>47
<211>84
<212>DNA
<213>Adenovirus type 12
<220>
<221>misc_feature
<223>Ad12 proximal pIX upstream sequence
<220>
<221>E1B 55K stopcodon
<222>(1)..(3)
<220>
<221>pIX startcodon
<222>(81)..(84)
<400>47
tgaggtaagt gggtggagct aggtgggatt ataaaaggct ggaagtcaac taaaaattgt 60
ttttgttctt ttaacagcac gatg 84
<210>48
<211>93
<212>DNA
<213>Adenovirus type 9
<220>
<221>misc_feature
<223>Ad9proximal pIX upstream sequence
<220>
<221>E1B 55K stopcodon
<222>(1)..(3)
<220>
<221>pIX startcodon
<222>(91)..(93)
<400>48
tagaggtagg tcgagtgagt agtgggcgtg gctaaggtga ctataaaggc gggtgtctta 60
cgagggtctt tttgcttttc tgcagacatc atg 93
<210>49
<211>73
<212>DNA
<213>Adenovirus type 40
<220>
<221>misc_feature
<223>Ad40proximal pIX upstream sequence
<220>
<221>E1B 55K stopcodon
<222>(1)..(3)
<220>
<221>pIX startcodon
<222>(71)..(73)
<400>49
taagggtaag gggcggagcc tattacaggt ataaaggttg gggtagagta aaaaaaaggg 60
aagttacaaa atg 73
<210>50
<211>90
<212>DNA
<213>Adenovirus type 4
<220>
<221>misc_feature
<223>Ad4proximal pIX upstream sequence
<220>
<221>E1B 55K stopcodon
<222>(1)..(3)
<220>
<221>pIX startcodon
<222>(88)..(90)
<400>50
tagagtgagt agtgttctgg ggcgggggag gacctgcatg agggccagaa taactgaaat 60
ctgtgctttt ctgtgtgttg cagcagcatg 90
<210>51
<211>90
<212>DNA
<213>simian Adenovirus type 25
<220>
<221>misc_feature
<223>sAd25 proximal pIX upstream sequence
<220>
<221>E1B 55K stopcodon
<222>(1)..(3)
<220>
<221>pIX startcodon
<222>(88)..(90)
<400>51
tagagtgagt agtgttctgg ggcgggggag gacctgcatg agggccagaa taactgaaat 60
ctgtgctttt ctgtgtgttg cagcagcatg 90
<210>52
<211>89
<212>DNA
<213>Adenovirus type 35
<220>
<221>misc_feature
<223>Ad35 proximal pIX upstream sequence
<220>
<221>E1B 55K stopcodon
<222>(1)..(3)
<220>
<221>pIX startcodon
<222>(87)..(89)
<400>52
taaggtgagt attgggaaaa ctttggggtg ggattttcag atggacagat tgagtaaaaa 60
tttgtttttt ctgtcttgca gctgacatg 89
<210>53
<211>89
<212>DNA
<213>Adenovirus type 11
<220>
<221>misc_feature
<223>Adll proximal pIX upstream sequence
<220>
<221>E1B 55K stopcodon
<222>(1)..(3)
<220>
<221>pIX stopcodon
<222>(87)..(89)
<400>53
taaggtgagt attgggaaaa ctttggggtg ggattttcag atggacagat tgagtaaaaa 60
tttgtttttt ctgtcttgca gctgtcatg 89
<210>54
<211>100
<212>DNA
<213>Adenovirus type 7
<220>
<221>misc_feature
<223>Ad7proximal pIX upstream sequence
<220>
<221>E1B 55K stopcodon
<222>(1)..(3)
<220>
<221>pIX startcodon
<222>(98)..(100)
<400>54
taaagtaagt agtgggggca aaatgtggat ggggactttc aggttggtaa ggtggacaaa 60
ttgggtaaat tttgttaatt tctgtcttgc agctgccatg 100
<210>55
<211>2430
<212>DNA
<213>Adenovirus type 35
<220>
<221>misc_feature
<223>Ad35E1B-pIX region
<400>55
aataaaaata tgttaactgt tcactggttt ttattgcttt ttgggcgggg actcaggtat 60
ataagtagaa gcagacctgt gtggttagct cataggagct ggctttcatc catggaggtt 120
tgggccattt tggaagacct taggaagact aggcaactgt tagagagcgc ttcggacgga 180
gtctccggtt tttggagatt ctggttcgct agtgaattag ctagggtagt ttttaggata 240
aaacaggact ataaacaaga atttgaaaag ttgttggtag attgcccagg actttttgaa 300
gctcttaatt tgggccatca ggttcacttt aaagaaaaag ttttatcagt tttagacttt 360
tcaaccccag gtagaactgc tgctgctgtg gcttttctta cttttatatt agataaatgg 420
atcccgcaga ctcatttcag caggggatac gttttggatt tcatagccac agcattgtgg 480
agaacatgga aggttcgcaa gatgaggaca atcttaggtt actggccagt gcagcctttg 540
ggtgtagcgg gaatcctgag gcatccaccg gtcatgccag cggttctgga ggaggaacag 600
caagaggaca acccgagagc cggcctggac cctccagtgg aggaggcgga gtagctgact 660
tgtctcctga actgcaacgg gtgcttactg gatctacgtc cactggacgg gataggggcg 720
ttaagaggga gagggcatcc agtggtactg atgctagatc tgagttggct ttaagtttaa 780
tgagtcgcag acgtcctgaa accatttggt ggcatgaggt tcagaaagag ggaagggatg 840
aagtttctgt attgcaggag aaatattcac tggaacaggt gaaaacatgt tggttggagc 900
cagaggatga ttgggcggtg gccattaaaa attatgccaa gatagctttg aggcctgata 960
aacagtataa gatcagtaga cggattaata tccggaatgc ttgttacata tctggaaatg 1020
gggctgaggt ggtaatagat actcaagaca agacagttat tagatgctgc atgatggata 1080
tgtggcctgg agtagtcggt atggaagcag tcacttttgt aaatgttaag tttaggggag 1140
atggttataa tggaatagtg tttatggcca ataccaaact tatattgcat ggttgtagct 1200
tttttggttt caacaatacc tgtgtagatg cctggggaca ggttagtgta cgggggtgta 1260
gtttctatgc gtgttggatt gccacagctg gcagaaccaa gagtcaattg tctctgaaga 1320
aatgcatatt ccaaagatgt aacctgggca ttctgaatga aggcgaagca agggtccgtc 1380
actgcgcttc tacagatact ggatgtttta ttttaattaa gggaaatgcc agcgtaaagc 1440
ataacatgat ttgtggtgct tccgatgaga ggccttatca aatgctcact tgtgctggtg 1500
ggcattgtaa tatgctggct actgtgcata ttgtttccca tcaacgcaaa aaatggcctg 1560
tttttgatca caatgtgttg accaagtgca ccatgcatgc aggtgggcgt agaggaatgt 1620
ttatgcctta ccagtgtaac atgaatcatg tgaaagtgtt gttggaacca gatgcctttt 1680
ccagaatgag cctaacagga atctttgaca tgaacacgca aatctggaag atcctgaggt 1740
atgatgatac gagatcgagg gtgcgcgcat gcgaatgcgg aggcaagcat gccaggttcc 1800
agccggtgtg tgtagatgtg accgaagatc tcagaccgga tcatttggtt attgcccgca 1860
ctggagcaga gttcggatcc agtggagaag aaactgacta aggtgagtat tgggaaaact 1920
ttggggtggg attttcagat ggacagattg agtaaaaatt tgttttttct gtcttgcagc 1980
tgacatgagt ggaaatgctt cttttaaggg gggagtcttc agcccttatc tgacagggcg 2040
tctcccatcc tgggcaggag ttcgtcagaa tgttatggga tctactgtgg atggaagacc 2100
cgttcaaccc gccaattctt caacgctgac ctatgctact ttaagttctt cacctttgga 2160
cgcagctgca gccgctgccg ccgcctctgt cgccgctaac actgtgcttg gaatgggtta 2220
ctatggaagc atcgtggcta attccacttc ctctaataac ccttctacac tgactcagga 2280
caagttactt gtccttttgg cccagctgga ggctttgacc caacgtctgg gtgaactttc 2340
tcagcaggtg gccgagttgc gagtacaaac tgagtctgct gtcggcacgg caaagtctaa 2400
ataaaaaaaa ttccagaatc aatgaataaa 2430
<210>56
<211>2429
<212>DNA
<213>Adenovirus type 11
<220>
<221>misc_feature
<223>Adll E1B-pIX region
<400>56
aataaaaata tgttaactgt tcactggttt ttattgcttt ttgggcgggg actcaggtat 60
ataagtagaa gcagacctgt gtggttagct cataggagct ggctttcatc catggaggtt 120
tgggccattt tggaagacct taggaagact aggcaactgt tagagaacgc ttcggacgga 180
gtctccggtt tttggagatt ctggttcgct agtgaattag ctagggtagt ttttaggata 240
aaacaggact ataaacaaga atttgaaaag ttgttggtag attgcccagg actttttgaa 300
gctcttaatt tgggccatca ggttcacttt aaagaaaaag ttttatcagt tttagacttt 360
tcaaccccag gtagaactgc tgctgctgtg gcttttctta cttttatatt agataaatgg 420
atcccgcaga ctcatttcag caggggatac gttttggatt tcatagccac agcattgtgg 480
agaacatgga aggttcgcaa gatgaggaca atcttaggtt actggccagt gcagcctttg 540
ggtgtagcgg gaatcctgag gcatccaccg gtcatgccag cggttctgga ggaggaacag 600
caagaggaca acccgagagc cggcctggac cctccagtgg aggaggcgga gtagctgact 660
tgtctcctga actgcaacgg gtgcttactg gatctacgtc cactggacgg gataggggcg 720
ttaagaggga gagggcatct agtggtactg atgctagatc tgagttggct ttaagtttaa 780
tgagtcgcag acgtcctgaa accatttggt ggcatgaggt tcagaaagag ggaagggatg 840
aagtttctgt attgcaggag aaatattcac tggaacaggt gaaaacatgt tggttggagc 900
ctgaggatga ttgggaggtg gccattaaaa attatgccaa gatagctttg aggcctgata 960
aacagtataa gattactaga cggattaata tccggaatgc ttgttacata tctggaaatg 1020
gggctgaggt ggtaatagat actcaagaca aggcagttat tagatgctgc atgatggata 1080
tgtggcctgg ggtagtcggt atggaagcag taacttttgt aaatgttaag tttaggggag 1140
atggttataa tggaatagtg tttatggcca ataccaaact tatattgcat ggttgtagct 1200
tttttggttt caacaatacc tgtgtagatg cctggggaca ggttagtgta cggggatgta 1260
gtttctatgc gtgttggatt gccacagctg gcagaaccaa gagtcaattg tctctgaaga 1320
aatgcatatt tcaaagatgt aacctgggca ttctgaatga aggcgaagca agggtccgcc 1380
actgcgcttc tacagatact ggatgtttta ttttgattaa gggaaatgcc agcgtaaagc 1440
ataacatgat ttgcggtgct tccgatgaga ggccttatca aatgctcact tgtgctggtg 1500
ggcattgtaa tatgctggct actgtgcata ttgtttccca tcaacgcaaa aaatggcctg 1560
tttttgatca caatgtgatg acgaagtgta ccatgcatgc aggtgggcgt agaggaatgt 1620
ttatgcctta ccagtgtaac atgaatcatg tgaaagtgtt gttggaacca gatgcctttt 1680
ccagaatgag ectaacagga atttttgaca tgaacatgca aatctggaag atcctgaggt 1740
atgatgatac gagatcgagg gtacgcgcat gcgaatgcgg aggcaagcat gccaggttcc 1800
agccggtgtg tgtagatgtg actgaagatc tcagaccgga tcatttggtt attgcccgca 1860
ctggagcaga gttcggatcc agtggagaag aaactgacta aggtgagtat tgggaaaact 1920
ttggggtggg attttcagat ggacagattg agtaaaaatt tgttttttct gtcttgcagc 1980
tgtcatgagt ggaaacgctt cttttaaggg gggagtcttc agcccttatc tgacagggcg 2040
tctcccatcc tgggcaggag ttcgtcagaa tgttatggga tctactgtgg atggaagacc 2100
cgtccaaccc gccaattctt caacgctgac ctatgctact ttaagttctt cacctttgga 2160
cgcagctgca gctgccgccg ccgcttctgt tgccgctaac actgtgcttg gaatgggtta 2220
ctatggaagc atcatggcta attccacttc ctctaataac ccttctaccc tgactcagga 2280
caagttactt gtccttttgg cccagctgga ggctttgacc caacgtctgg gtgaactttc 2340
tcagcaggtg gtcgagttgc gagtacaaac tgagtctgct gtcggcacgg caaagtctaa 2400
ataaaaaaat cccagaatca atgaataaa 2429
<210>57
<211>2426
<212>DNA
<213>Adenovirus type 7
<220>
<221>misc_feature
<223>Ad7 E1B-pIX region
<400>57
aataaaatta tgtcagctgc tgagtgtttt attacttctt gggtggggtc ttggatatat 60
aagtaggagc agatctgtgt ggttagctca cagcaacttg ctgccatcca tggaggtttg 120
ggctatcttg gaagacctca gacagactag gctactacta gaaaacgcct cggacggagt 180
ctctggcctt tggagattct ggttcggtgg tgatctagct aggctagtgt ttaggataaa 240
acaggactac agggaagaat ttgaaaagtt attggacgac attccaggac tttttgaagc 300
tcttaacttg ggccatcagg ctcattttaa ggagaaggtt ttatcagttt tagatttttc 360
tactcctggt agaactgctg ctgctgtagc ttttcttact tttatattgg ataaatggat 420
ccgccaaact cacttcagca agggatacgt tttggatttc atagcagcag ctttgtggag 480
aacatggaag gctcgcagga tgaggacaat cttagattac tggccagtgc agcctctggg 540
agtagcaggg atactgagac acccaccgac catgccagcg gttctgcagg aggagcagca 600
ggaggacaat ccgagagccg gcctggaccc tccggtggag gagtagctga cctgtttcct 660
gaactgcgac gggtgcttac taggtctacg accagtggac agaacagggg aattaagagg 720
gagaggaatc ctagtgggaa taattcaaga accgagttgg ctttaagttt aatgagccgc 780
aggcgtcctg aaactgtttg gtggcatgag gttcagagcg aaggcaggga tgaagtttca 840
atattgcagg agaaatattc actagaacaa cttaagacct gttggttgga acctgaggat 900
gattgggagg tggccattag gaattatgct aagatatctc tgaggcctga taaacaatat 960
agaattacta agaagattaa tattagaaat gcatgctaca tatcagggaa tggggcagag 1020
gttataatag atacacaaga taaagcagct tttagatgtt gtatgatggg tatgtggcca 1080
ggggttgtcg gcatggaagc aataacactt atgaatatta ggtttagagg ggatgggtat 1140
aatggcattg tatttatggc taacactaag ctgattctac atggttgtag cttttttggg 1200
tttaataata cgtgtgtaga agcttggggg caagttagtg tgaggggttg tagtttttat 1260
gcatgctgga ttgcaacatc aggtagggtg aagagtcagt tgtctgtgaa gaaatgcatg 1320
tttgagagat gtaatcttgg catactgaat gaaggtgaag caagggtccg ccactgcgca 1380
gctacagaaa ctgcctgctt cattctaata aagggaaatg ccagtgtgaa gcataatatg 1440
atctgtggac attcggatga gaggccttat cagatgctaa cctgcgctgg tggacattgc 1500
aatattcttg ctaccgtgca tatcgtttca catgcacgca agaaatggcc tgtatttgaa 1560
cataatgtga ttaccaagtg caccatgcat ataggtggtc gcaggggaat gtttatgcct 1620
taccagtgta acatgaatca tgtgaaggta atgttggaac cagatgcctt ttccagagtg 1680
agcgtaacag gaatctttga tatgaatatt caactatgga agatcctgag atatgatgac 1740
actaaaccaa gggtgcgcgc atgcgaatgc ggaggcaagc atgctagatt ccagccggtg 1800
tgcgtggatg tgactgaaga cctgaggccc gatcatttgg tgcttgcctg cactggagcg 1860
gagttcggtt ctagtggtga agaaactgac taaagtaagt agtgggggca aaatgtggat 1920
ggggactttc aggttggtaa ggtggacaaa ttgggtaaat tttgttaatt tctgtcttgc 1980
agctgccatg agtggaagcg cttcttttga ggggggagta tttagccctt atctgacggg 2040
caggctccca ccatgggcag gagttcgtca gaatgtcatg ggatccactg tggatgggag 2100
acccgtccag cccgccaatt cctcaacgct gacctatgcc actttgagtt cgtcaccatt 2160
ggatgcagct gcagccgccg ccgctactgc tgccgccaac accatccttg gaatgggcta 2220
ttacggaagc attgttgcca attccagttc ctctaataat ccttcaaccc tggctgagga 2280
caagctactt gttctcttgg ctcagctcga ggccttaacc caacgcttag gcgaactgtc 2340
taagcaggtg gcccagttgc gtgagcaaac tgagtctgct gttgccacag caaagtctaa 2400
ataaagatct caaatcaata aataaa 2426
<210>58
<211>240
<212>DNA
<213>Adenovirus type 11
<220>
<221>misc_feature
<223>Adll pIX cDNA sequence
<400>58
cgactggagc acgaggacac tgacatggac tgaaggagta gaaatcattt ggttattgcc 60
cgcactggag cagagttcgg atccagtgga gaagaaactg actaagctgt catgagtgga 120
aacgcttctt ttaagggggg agtcttcagc ccttatctga cagggcgtct cccatcctgg 180
gcaggagttc gtcagaatgt tatgggatct actgtggatg gaacacccgt tcaacccgcc 240
<210>59
<211>227
<212>DNA
<213>Adenovirus type 35
<220>
<221>misc_feature
<223>Ad35 pIX cDNA sequence
<400>59
ggacactgac atggactgaa ggagtagaaa atcatttggt tattgcccgc actggagcag 60
agttcggatc cagtggagaa gaaactgact aagctgacat gagtggaaat gcttctttta 120
aggggggagt cttcagccct tatctgacag ggcgtctccc atcctgggca ggagttcgtc 180
agaatgttat gggatctact gtggatggaa gacccgttca acccgcc 227
<210>60
<211>289
<212>DNA
<213>Adenovirus type 35
<220>
<221>misc_feature
<223>wild-type Ad35 sequence nt 3339-3628
<220>
<221>Intron
<222>(62)..(138)
<220>
<221>startcodon pIX
<222>(146)..(148)
<400>60
atcatttggt tattgcccgc actggagcag agttcggatc cagtggagaa gaaactgact 60
aaggtgagta ttgggaaaac tttggggtgg gattttcaga tggacagatt gagtaaaaat 120
ttgttttttc tgtcttgcag ctgacatgag tggaaatgct tcttttaagg ggggagtctt 180
cagcccttat ctgacagggc gtctcccatc ctgggcagga gttcgtcaga atgttatggg 240
atctactgtg gatggaagac ccgttcaacc cgccaattct tcaacgctg 289
<210>61
<211>294
<212>PRT
<213>Adenovirus type 5
<220>
<221>MISC_FEATURE
<223>amino acid sequence of the E4-orf6 protein of Ad5
<400>61
Met Thr Thr Ser Gly Val Pro Phe Gly Met Thr Leu Arg Pro Thr Arg
1 5 10 15
Ser Arg Leu Ser Arg Arg Thr Pro Tyr Ser Arg Asp Arg Leu Pro Pro
20 25 30
Phe Glu Thr Glu Thr Arg Ala Thr Ile Leu Glu Asp His Pro Leu Leu
35 40 45
Pro Glu Cys Asn Thr Leu Thr Met His Asn Val Ser Tyr Val Arg Gly
50 55 60
Leu Pro Cys Ser Val Gly Phe Thr Leu Ile Gln Glu Trp Val Val Pro
65 70 75 80
Trp Asp Met Val Leu Thr Arg Glu Glu Leu Val Ile Leu Arg Lys Cys
85 90 95
Met His Val Cys Leu Cys Cys Ala Asn Ile Asp Ile Met Thr Ser Met
100 105 110
Met Ile His Gly Tyr Glu Ser Trp Ala Leu His Cys His Cys Ser Ser
115 120 125
Pro Gly Ser Leu Gln Cys Ile Ala Gly Gly Gln Val Leu Ala Ser Trp
130 135 140
Phe Arg Met Val Val Asp Gly Ala Met Phe Asn Gln Arg Phe Ile Trp
145 150 155
Tyr Arg Glu Val Val Asn Tyr Asn Met Pro Lys Glu Val Met Phe Met
165 170 175
Ser Ser Val Phe Met Arg Gly Arg His Leu Ile Tyr Leu Arg Leu Trp
180 185 190
Tyr Asp Gly His Val Gly Ser Val Val Pro Ala Met Ser Phe Gly Tyr
195 200 205
Ser Ala Leu His Cys Gly Ile Leu Asn Asn Ile Val Val Leu Cys Cys
210 215 220
Ser Tyr Cys Ala Asp Leu Ser Glu Ile Arg Val Arg Cys Cys Ala Arg
225 230 240
Arg Thr Arg Arg Leu Met Leu Arg Ala Val Arg Ile Ile Ala Glu Glu
245 250 255
Thr Thr Ala Met Leu Tyr Ser Cys Arg Thr Glu Arg Arg Arg Gln Gln
260 265 270
Phe Ile Arg Ala Leu Leu Gln His His Arg Pro Ile Leu Met His Asp
275 280 285
Tyr Asp Ser Thr Pro Met
290
<210>62
<211>299
<212>PRT
<213>Adenovirus type 35
<220>
<221>MISC_FEATURE
<223>amino acid sequence of the E4-orf6 protein of Ad35
<400>62
Met Ser Gly Ser Asn Ser Ile Met Thr Arg Leu Arg Ala Arg Ser Thr
1 5 10 15
Ser Cys Ala Arg His His Pro Tyr Thr Arg Ala Gln Leu Pro Arg Cys
20 25 30
Glu Glu Asn Glu Thr Arg Ala Ser Met Thr Glu Asp His Pro Leu Leu
35 40 45
Pro Asp Cys Asp Thr Met Thr Met His Ser Val Ser Cys Val Arg Gly
50 55 60
Leu Pro Cys Ser Ala Ser Phe Thr Val Leu Gln Glu Leu Pro Ile Pro
65 70 75 80
Trp Asp Met Phe Leu Asn Pro Glu Glu Leu Lys Ile Met Arg Arg Cys
85 90 95
Met His Leu Cys Leu Cys Cys Ala Thr Ile Asp Ile Phe His Ser Gln
100 105 110
Val Ile His Gly Arg Glu Asn Trp Val Leu His Cys His Cys Asn Gln
115 120 125
Gln Gly Ser Leu Gln Cys Met Ala Gly Gly Ala Val Leu Ala Val Trp
130 135 140
Phe Arg Lys Val Ile Leu Gly Cys Met Ile Asn Gln Arg Cys Pro Trp
145 150 155 160
Tyr Arg Gln Ile Val Asn Met His Met Pro Lys Glu Ile Met Tyr Val
165 170 175
Gly Ser Val Phe Leu Arg Glu Arg His Leu Ile Tyr Ile Lys Leu Trp
180 185 190
Tyr Asp Gly His Ala Gly Ala Ile Ile Ser Asp Met Ser Phe Gly Trp
195 200 205
Ser Ala Phe Asn Tyr Gly Leu Leu Asn Asn Ile Val Ile Met Cys Cys
210 215 220
Thr Tyr Cys Lys Asp Leu Ser Glu Ile Arg Met Arg Cys Cys Ala His
225 230 235 240
Arg Thr Arg Lys Leu Met Leu Arg Ala Ile Lys Ile Met Leu Gln Glu
245 250 255
Thr Val Asp Pro Asp Pro Ile Asn Ser Ser Arg Thr Glu Arg Arg Arg
260 265 270
Gln Arg Leu Leu Val Gly Leu Met Arg His Asn Arg Pro Ile Pro Phe
275 280 285
Ser Asp Tyr Asp Ser His Arg Ser Ser Ser Arg
290 295
<210>63
<211>150
<212>PRT
<213>Adenovirus type 5
<220>
<221>misc feature
<223>amino acid sequence of the E4-orf6+7 protein of Ad5
<400>63
Met Thr Thr Ser Gly Val Pro Phe Gly Met Thr Leu Arg Pro Thr Arg
1 5 10 15
Ser Arg Leu Ser Arg Arg Thr Pro Tyr Ser Arg Asp Arg Leu Pro Pro
20 25 30
Phe Glu Thr Glu Thr Arg Ala Thr Ile Leu Glu Asp His Pro Leu Leu
35 40 45
Pro Glu Cys Asn Thr Leu Thr Met His Asn Ala Trp Thr Ser Pro Ser
50 55 60
Pro Pro Val Lys Gln Pro Gln Val Gly Gln Gln Pro Val Ala Gln Gln
65 70 75 80
Leu Asp Ser Asp Met Asn Leu Ser Glu Leu Pro Gly Glu Phe Ile Asn
85 90 95
Ile Thr Asp Glu Arg Leu Ala Arg Gln Glu Thr Val Trp Asn Ile Thr
100 105 110
Pro Lys Asn Met Ser Val Thr His Asp Met Met Leu Phe Lys Ala Ser
115 120 125
Arg Gly Glu Arg Thr Val Tyr Ser Val Cys Trp Glu Gly Gly Gly Arg
130 135 140
Leu Asn Thr Arg Val Leu
145 150
<210>64
<211>141
<212>PRT
<213>Adenovirus type 35
<220>
<221>misc_feature
<223>amino acid sequence of the E4-orf6+7 protein of Ad35
<400>64
Met Ser Gly Ser Asn Ser Ile Met Thr Arg Leu Arg Ala Arg Ser Thr
1 5 10 15
Ser Cys Ala Arg His His Pro Tyr Thr Arg Ala Gln Leu Pro Arg Cys
20 25 30
Glu Glu Asn Glu Thr Arg Ala Ser Met Thr Glu Asp His Pro Leu Leu
35 40 45
Pro Asp Cys Asp Thr Met Thr Met His Ser Met Thr Val Ile Gln Thr
50 55 60
Pro Glu Ser His Pro Gln Gln Leu Asp Cys Glu Ser Ala Leu Lys Asp
65 70 75 80
Tyr Arg Asp Gly Phe Leu Ser Ile Thr Asp Pro Arg Leu Ala Arg Ser
85 90 95
Glu Thr Val Trp Asn Val Glu Ser Lys Thr Met Ser Ile Ser Asn Gly
100 105 110
Ile Gln Met Phe Lys Ala Val Arg Gly Glu Arg Leu Val Tyr Ser Val
115 120 125
Lys Trp Glu Gly Gly Gly Lys Ile Thr Thr Arg Ile Leu
130 135 140
<210>65
<211>150
<212>PRT
<213>Artificial
<220>
<223>amino acid sequence of the E4-orf6+7 fusion protein from Ad5 and
Ad35
<400>65
Met Thr Thr Ser Gly Val Pro Phe Gly Met Thr Leu Arg Pro Thr Arg
1 5 10 15
Ser Arg Leu Ser Arg Arg Thr Pro Tyr Ser Arg Asp Arg Leu Pro Pro
20 25 30
Phe Glu Thr Glu Thr Arg Ala Thr Ile Leu Glu Asp His Pro Leu Leu
35 40 45
Pro Glu Cys Asn Thr Leu Thr Met His Asn Ala Trp Thr Ser Pro Ser
50 55 60
Pro Pro Val Lys Gln Pro Gln Val Gly Gln Gln Pro Val Ala Gln Gln
65 70 75 80
Leu Asp Ser Asp Met Asn Leu Ser Glu Leu Pro Gly Glu Phe Ile Asn
85 90 95
Ile Thr Asp Glu Arg Leu Ala Arg Gln Glu Thr Val Trp Asn Ile Thr
100 105 110
Pro Lys Asn Met Ser Val Thr His Asp Met Met Leu Phe Lys Ala Ser
115 120 125
Arg Gly Glu Arg Thr Val Tyr Ser Val Lys Trp Glu Gly Gly Gly Lys
130 135 140
Ile Thr Thr Arg Ile Leu
145 150