JP5026958B2 - リコンビナーゼポリメラーゼ増幅 - Google Patents
リコンビナーゼポリメラーゼ増幅 Download PDFInfo
- Publication number
- JP5026958B2 JP5026958B2 JP2007514201A JP2007514201A JP5026958B2 JP 5026958 B2 JP5026958 B2 JP 5026958B2 JP 2007514201 A JP2007514201 A JP 2007514201A JP 2007514201 A JP2007514201 A JP 2007514201A JP 5026958 B2 JP5026958 B2 JP 5026958B2
- Authority
- JP
- Japan
- Prior art keywords
- dna
- recombinase
- polymerase
- primer
- amplification
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
- 102000018120 Recombinases Human genes 0.000 title claims abstract description 259
- 108010091086 Recombinases Proteins 0.000 title claims abstract description 259
- 238000003199 nucleic acid amplification method Methods 0.000 title claims abstract description 225
- 230000003321 amplification Effects 0.000 title claims abstract description 224
- 238000006243 chemical reaction Methods 0.000 claims abstract description 332
- 238000000034 method Methods 0.000 claims abstract description 232
- 108020004414 DNA Proteins 0.000 claims abstract description 168
- 102000053602 DNA Human genes 0.000 claims abstract description 154
- 108090000623 proteins and genes Proteins 0.000 claims abstract description 107
- 102000004169 proteins and genes Human genes 0.000 claims abstract description 99
- 102000004190 Enzymes Human genes 0.000 claims abstract description 48
- 108090000790 Enzymes Proteins 0.000 claims abstract description 48
- 102000016928 DNA-directed DNA polymerase Human genes 0.000 claims abstract description 45
- 108010014303 DNA-directed DNA polymerase Proteins 0.000 claims abstract description 45
- 150000007523 nucleic acids Chemical class 0.000 claims description 172
- 102000039446 nucleic acids Human genes 0.000 claims description 168
- 108020004707 nucleic acids Proteins 0.000 claims description 168
- ZKHQWZAMYRWXGA-KQYNXXCUSA-N Adenosine triphosphate Chemical group C1=NC=2C(N)=NC=NC=2N1[C@@H]1O[C@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)[C@@H](O)[C@H]1O ZKHQWZAMYRWXGA-KQYNXXCUSA-N 0.000 claims description 163
- 230000000694 effects Effects 0.000 claims description 155
- 101800001690 Transmembrane protein gp41 Proteins 0.000 claims description 141
- 101800001466 Envelope glycoprotein E1 Proteins 0.000 claims description 136
- 241000588724 Escherichia coli Species 0.000 claims description 101
- 235000018102 proteins Nutrition 0.000 claims description 98
- 108010055016 Rec A Recombinases Proteins 0.000 claims description 93
- 102000001218 Rec A Recombinases Human genes 0.000 claims description 93
- 229920001223 polyethylene glycol Polymers 0.000 claims description 65
- 239000002202 Polyethylene glycol Substances 0.000 claims description 59
- 239000000203 mixture Substances 0.000 claims description 51
- 102000052510 DNA-Binding Proteins Human genes 0.000 claims description 50
- 239000003153 chemical reaction reagent Substances 0.000 claims description 49
- 108060004795 Methyltransferase Proteins 0.000 claims description 45
- NLTUCYMLOPLUHL-KQYNXXCUSA-N adenosine 5'-[gamma-thio]triphosphate Chemical compound C1=NC=2C(N)=NC=NC=2N1[C@@H]1O[C@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=S)[C@@H](O)[C@H]1O NLTUCYMLOPLUHL-KQYNXXCUSA-N 0.000 claims description 39
- 239000012634 fragment Substances 0.000 claims description 39
- 101100048967 Enterobacteria phage T4 uvsY gene Proteins 0.000 claims description 33
- 101710176276 SSB protein Proteins 0.000 claims description 30
- 101710126859 Single-stranded DNA-binding protein Proteins 0.000 claims description 30
- 101710116602 DNA-Binding protein G5P Proteins 0.000 claims description 27
- 101710162453 Replication factor A Proteins 0.000 claims description 27
- 101710176758 Replication protein A 70 kDa DNA-binding subunit Proteins 0.000 claims description 27
- 239000000376 reactant Substances 0.000 claims description 25
- -1 morpholino backbone nucleic acid Chemical class 0.000 claims description 23
- DRBBFCLWYRJSJZ-UHFFFAOYSA-N N-phosphocreatine Chemical compound OC(=O)CN(C)C(=N)NP(O)(O)=O DRBBFCLWYRJSJZ-UHFFFAOYSA-N 0.000 claims description 22
- 108010017826 DNA Polymerase I Proteins 0.000 claims description 21
- 102000004594 DNA Polymerase I Human genes 0.000 claims description 21
- 108091008324 binding proteins Proteins 0.000 claims description 20
- 230000008929 regeneration Effects 0.000 claims description 18
- 238000011069 regeneration method Methods 0.000 claims description 18
- 102000016559 DNA Primase Human genes 0.000 claims description 15
- 108010092681 DNA Primase Proteins 0.000 claims description 15
- 239000000872 buffer Substances 0.000 claims description 15
- 150000001875 compounds Chemical class 0.000 claims description 13
- 241000549435 Pria Species 0.000 claims description 12
- SCVFZCLFOSHCOH-UHFFFAOYSA-M potassium acetate Chemical compound [K+].CC([O-])=O SCVFZCLFOSHCOH-UHFFFAOYSA-M 0.000 claims description 12
- 102000004420 Creatine Kinase Human genes 0.000 claims description 10
- 108010042126 Creatine kinase Proteins 0.000 claims description 10
- OAKPWEUQDVLTCN-NKWVEPMBSA-N 2',3'-Dideoxyadenosine-5-triphosphate Chemical compound C1=NC=2C(N)=NC=NC=2N1[C@H]1CC[C@@H](CO[P@@](O)(=O)O[P@](O)(=O)OP(O)(O)=O)O1 OAKPWEUQDVLTCN-NKWVEPMBSA-N 0.000 claims description 9
- 235000014469 Bacillus subtilis Nutrition 0.000 claims description 9
- 239000008351 acetate buffer Substances 0.000 claims description 9
- XPPKVPWEQAFLFU-UHFFFAOYSA-J diphosphate(4-) Chemical compound [O-]P([O-])(=O)OP([O-])([O-])=O XPPKVPWEQAFLFU-UHFFFAOYSA-J 0.000 claims description 9
- 235000011180 diphosphates Nutrition 0.000 claims description 9
- UEGPKNKPLBYCNK-UHFFFAOYSA-L magnesium acetate Chemical compound [Mg+2].CC([O-])=O.CC([O-])=O UEGPKNKPLBYCNK-UHFFFAOYSA-L 0.000 claims description 9
- PIEPQKCYPFFYMG-UHFFFAOYSA-N tris acetate Chemical compound CC(O)=O.OCC(N)(CO)CO PIEPQKCYPFFYMG-UHFFFAOYSA-N 0.000 claims description 9
- 244000063299 Bacillus subtilis Species 0.000 claims description 8
- 239000007850 fluorescent dye Substances 0.000 claims description 8
- 229920002534 Polyethylene Glycol 1450 Polymers 0.000 claims description 7
- 239000011654 magnesium acetate Substances 0.000 claims description 7
- 235000011285 magnesium acetate Nutrition 0.000 claims description 7
- 229940069446 magnesium acetate Drugs 0.000 claims description 7
- 101150103919 recO gene Proteins 0.000 claims description 7
- 101150033993 recR gene Proteins 0.000 claims description 7
- 108010061982 DNA Ligases Proteins 0.000 claims description 6
- 102000012410 DNA Ligases Human genes 0.000 claims description 6
- XGAOYVYIONYFMI-VWJVIAGJSA-N [(2r,3s,4r,5r)-5-(6-aminopurin-9-yl)-3,4-dihydroxyoxolan-2-yl]methyl [hydroxy(phosphonooxy)phosphinothioyl] hydrogen phosphate Chemical compound C1=NC=2C(N)=NC=NC=2N1[C@@H]1O[C@H](COP(O)(=O)OP(O)(=S)OP(O)(O)=O)[C@@H](O)[C@H]1O XGAOYVYIONYFMI-VWJVIAGJSA-N 0.000 claims description 6
- 235000011056 potassium acetate Nutrition 0.000 claims description 6
- 238000006467 substitution reaction Methods 0.000 claims description 6
- 239000004475 Arginine Substances 0.000 claims description 5
- 108010071146 DNA Polymerase III Proteins 0.000 claims description 5
- 102000007528 DNA Polymerase III Human genes 0.000 claims description 5
- 241000193385 Geobacillus stearothermophilus Species 0.000 claims description 5
- 229910019142 PO4 Inorganic materials 0.000 claims description 5
- 235000001014 amino acid Nutrition 0.000 claims description 5
- ODKSFYDXXFIFQN-UHFFFAOYSA-N arginine Natural products OC(=O)C(N)CCCNC(N)=N ODKSFYDXXFIFQN-UHFFFAOYSA-N 0.000 claims description 5
- 239000010452 phosphate Substances 0.000 claims description 5
- 230000000087 stabilizing effect Effects 0.000 claims description 5
- 239000004472 Lysine Substances 0.000 claims description 4
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 claims description 4
- LTGPFZWZZNUIIK-LURJTMIESA-N Lysol Chemical compound NCCCC[C@H](N)CO LTGPFZWZZNUIIK-LURJTMIESA-N 0.000 claims description 4
- 102000009609 Pyrophosphatases Human genes 0.000 claims description 4
- 108010009413 Pyrophosphatases Proteins 0.000 claims description 4
- 239000003638 chemical reducing agent Substances 0.000 claims description 4
- 239000002532 enzyme inhibitor Substances 0.000 claims description 4
- NBIIXXVUZAFLBC-UHFFFAOYSA-K phosphate Chemical compound [O-]P([O-])([O-])=O NBIIXXVUZAFLBC-UHFFFAOYSA-K 0.000 claims description 4
- 101100117496 Sulfurisphaera ohwakuensis pol-alpha gene Proteins 0.000 claims description 3
- 108010063113 DNA Polymerase II Proteins 0.000 claims description 2
- 102000010567 DNA Polymerase II Human genes 0.000 claims description 2
- 108010001132 DNA Polymerase beta Proteins 0.000 claims description 2
- 102000001996 DNA Polymerase beta Human genes 0.000 claims description 2
- 101100443914 Enterobacteria phage T4 43 gene Proteins 0.000 claims description 2
- 101000906736 Escherichia phage Mu DNA circularization protein N Proteins 0.000 claims description 2
- 229920002594 Polyethylene Glycol 8000 Polymers 0.000 claims description 2
- 101000764570 Streptomyces phage phiC31 Probable tape measure protein Proteins 0.000 claims description 2
- AYFVYJQAPQTCCC-UHFFFAOYSA-N Threonine Natural products CC(O)C(N)C(O)=O AYFVYJQAPQTCCC-UHFFFAOYSA-N 0.000 claims description 2
- 239000004473 Threonine Substances 0.000 claims description 2
- 235000004279 alanine Nutrition 0.000 claims description 2
- 125000000637 arginyl group Chemical group N[C@@H](CCCNC(N)=N)C(=O)* 0.000 claims description 2
- 229940125532 enzyme inhibitor Drugs 0.000 claims description 2
- ZDXPYRJPNDTMRX-UHFFFAOYSA-N glutamine Natural products OC(=O)C(N)CCC(N)=O ZDXPYRJPNDTMRX-UHFFFAOYSA-N 0.000 claims description 2
- 239000003550 marker Substances 0.000 claims description 2
- 101710197907 rDNA transcriptional regulator pol5 Proteins 0.000 claims description 2
- 230000002285 radioactive effect Effects 0.000 claims description 2
- 102000023732 binding proteins Human genes 0.000 claims 3
- 150000001413 amino acids Chemical class 0.000 claims 2
- 125000003588 lysine group Chemical group [H]N([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])(N([H])[H])C(*)=O 0.000 claims 2
- 101900234631 Escherichia coli DNA polymerase I Proteins 0.000 claims 1
- QNAYBMKLOCPYGJ-REOHCLBHSA-N L-alanine Chemical compound C[C@H](N)C(O)=O QNAYBMKLOCPYGJ-REOHCLBHSA-N 0.000 claims 1
- ZDXPYRJPNDTMRX-VKHMYHEASA-N L-glutamine Chemical compound OC(=O)[C@@H](N)CCC(N)=O ZDXPYRJPNDTMRX-VKHMYHEASA-N 0.000 claims 1
- AYFVYJQAPQTCCC-GBXIJSLDSA-N L-threonine Chemical compound C[C@@H](O)[C@H](N)C(O)=O AYFVYJQAPQTCCC-GBXIJSLDSA-N 0.000 claims 1
- 150000001484 arginines Chemical group 0.000 claims 1
- 108091034117 Oligonucleotide Proteins 0.000 abstract description 282
- 230000006798 recombination Effects 0.000 abstract description 78
- 238000005215 recombination Methods 0.000 abstract description 78
- 239000000523 sample Substances 0.000 abstract description 76
- 238000013459 approach Methods 0.000 abstract description 66
- 230000008901 benefit Effects 0.000 abstract description 26
- 238000012544 monitoring process Methods 0.000 abstract description 11
- 230000037452 priming Effects 0.000 abstract description 11
- 238000001502 gel electrophoresis Methods 0.000 abstract description 8
- 238000011109 contamination Methods 0.000 abstract 1
- 239000013615 primer Substances 0.000 description 320
- 230000015572 biosynthetic process Effects 0.000 description 140
- 238000003786 synthesis reaction Methods 0.000 description 118
- 239000000047 product Substances 0.000 description 109
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 description 101
- 108020004682 Single-Stranded DNA Proteins 0.000 description 89
- 239000000306 component Substances 0.000 description 84
- 101150079601 recA gene Proteins 0.000 description 66
- 238000003752 polymerase chain reaction Methods 0.000 description 62
- 238000002474 experimental method Methods 0.000 description 59
- 230000010076 replication Effects 0.000 description 53
- 102000011931 Nucleoproteins Human genes 0.000 description 52
- 108010061100 Nucleoproteins Proteins 0.000 description 52
- 230000000295 complement effect Effects 0.000 description 49
- 229940088598 enzyme Drugs 0.000 description 46
- 239000012071 phase Substances 0.000 description 43
- 239000000543 intermediate Substances 0.000 description 42
- 238000006460 hydrolysis reaction Methods 0.000 description 40
- 239000002773 nucleotide Substances 0.000 description 40
- 125000003729 nucleotide group Chemical group 0.000 description 40
- 238000009396 hybridization Methods 0.000 description 39
- 230000007062 hydrolysis Effects 0.000 description 38
- 230000006399 behavior Effects 0.000 description 37
- 230000006870 function Effects 0.000 description 34
- 230000009545 invasion Effects 0.000 description 34
- 102100023038 WD and tetratricopeptide repeats protein 1 Human genes 0.000 description 32
- 230000027455 binding Effects 0.000 description 31
- 230000015556 catabolic process Effects 0.000 description 31
- 238000006731 degradation reaction Methods 0.000 description 31
- 230000008685 targeting Effects 0.000 description 31
- 230000006820 DNA synthesis Effects 0.000 description 29
- 238000001514 detection method Methods 0.000 description 29
- 108091093088 Amplicon Proteins 0.000 description 28
- 238000002844 melting Methods 0.000 description 28
- 230000008018 melting Effects 0.000 description 28
- 238000011068 loading method Methods 0.000 description 25
- 108060002716 Exonuclease Proteins 0.000 description 24
- 102000013165 exonuclease Human genes 0.000 description 24
- 230000003993 interaction Effects 0.000 description 24
- 238000000338 in vitro Methods 0.000 description 23
- 239000000243 solution Substances 0.000 description 23
- 108700020911 DNA-Binding Proteins Proteins 0.000 description 21
- 230000007246 mechanism Effects 0.000 description 21
- 238000013508 migration Methods 0.000 description 21
- 230000005012 migration Effects 0.000 description 21
- 230000008569 process Effects 0.000 description 21
- 238000006073 displacement reaction Methods 0.000 description 20
- 230000004544 DNA amplification Effects 0.000 description 19
- 108010025076 Holoenzymes Proteins 0.000 description 19
- 230000007423 decrease Effects 0.000 description 19
- 230000001404 mediated effect Effects 0.000 description 19
- 230000035945 sensitivity Effects 0.000 description 19
- 238000009825 accumulation Methods 0.000 description 18
- 238000007792 addition Methods 0.000 description 18
- OPTASPLRGRRNAP-UHFFFAOYSA-N cytosine Chemical compound NC=1C=CNC(=O)N=1 OPTASPLRGRRNAP-UHFFFAOYSA-N 0.000 description 18
- 102000014914 Carrier Proteins Human genes 0.000 description 17
- 230000001965 increasing effect Effects 0.000 description 17
- 108700014590 single-stranded DNA binding proteins Proteins 0.000 description 17
- 235000000346 sugar Nutrition 0.000 description 17
- 230000001419 dependent effect Effects 0.000 description 16
- 239000007795 chemical reaction product Substances 0.000 description 15
- 238000010790 dilution Methods 0.000 description 15
- 239000012895 dilution Substances 0.000 description 15
- 230000033001 locomotion Effects 0.000 description 15
- 101710163270 Nuclease Proteins 0.000 description 14
- 239000000539 dimer Substances 0.000 description 14
- 241000894007 species Species 0.000 description 14
- 239000003155 DNA primer Substances 0.000 description 13
- 230000009471 action Effects 0.000 description 13
- 238000004458 analytical method Methods 0.000 description 13
- 239000003795 chemical substances by application Substances 0.000 description 13
- 239000000758 substrate Substances 0.000 description 13
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N (+)-Biotin Chemical compound N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 description 12
- 241000701533 Escherichia virus T4 Species 0.000 description 12
- 108091028043 Nucleic acid sequence Proteins 0.000 description 12
- 238000003556 assay Methods 0.000 description 12
- 238000010494 dissociation reaction Methods 0.000 description 12
- 230000005593 dissociations Effects 0.000 description 12
- 239000000499 gel Substances 0.000 description 12
- PCHJSUWPFVWCPO-UHFFFAOYSA-N gold Chemical compound [Au] PCHJSUWPFVWCPO-UHFFFAOYSA-N 0.000 description 12
- 229910052737 gold Inorganic materials 0.000 description 12
- 239000010931 gold Substances 0.000 description 12
- 238000011534 incubation Methods 0.000 description 12
- 239000013616 RNA primer Substances 0.000 description 11
- 239000000975 dye Substances 0.000 description 11
- 238000001727 in vivo Methods 0.000 description 11
- 230000000977 initiatory effect Effects 0.000 description 11
- 239000000178 monomer Substances 0.000 description 11
- 239000011541 reaction mixture Substances 0.000 description 11
- 230000002829 reductive effect Effects 0.000 description 11
- 230000002441 reversible effect Effects 0.000 description 11
- 230000004568 DNA-binding Effects 0.000 description 10
- NYHBQMYGNKIUIF-UUOKFMHZSA-N Guanosine Chemical compound C1=NC=2C(=O)NC(N)=NC=2N1[C@@H]1O[C@H](CO)[C@@H](O)[C@H]1O NYHBQMYGNKIUIF-UUOKFMHZSA-N 0.000 description 10
- 238000013461 design Methods 0.000 description 10
- 238000000926 separation method Methods 0.000 description 10
- 239000000126 substance Substances 0.000 description 10
- RWQNBRDOKXIBIV-UHFFFAOYSA-N thymine Chemical compound CC1=CNC(=O)NC1=O RWQNBRDOKXIBIV-UHFFFAOYSA-N 0.000 description 10
- 239000001226 triphosphate Substances 0.000 description 10
- 235000011178 triphosphate Nutrition 0.000 description 10
- 238000000137 annealing Methods 0.000 description 9
- 230000001976 improved effect Effects 0.000 description 9
- 238000004519 manufacturing process Methods 0.000 description 9
- 238000012986 modification Methods 0.000 description 9
- 230000004048 modification Effects 0.000 description 9
- 239000002777 nucleoside Substances 0.000 description 9
- 108090000765 processed proteins & peptides Proteins 0.000 description 9
- 229920003987 resole Polymers 0.000 description 9
- 238000012360 testing method Methods 0.000 description 9
- 229940104302 cytosine Drugs 0.000 description 8
- 230000035772 mutation Effects 0.000 description 8
- 238000012545 processing Methods 0.000 description 8
- 108091008146 restriction endonucleases Proteins 0.000 description 8
- 239000011616 biotin Substances 0.000 description 7
- 229960002685 biotin Drugs 0.000 description 7
- 230000002255 enzymatic effect Effects 0.000 description 7
- 230000006911 nucleation Effects 0.000 description 7
- 238000010899 nucleation Methods 0.000 description 7
- 230000009467 reduction Effects 0.000 description 7
- 241000894006 Bacteria Species 0.000 description 6
- UOLVQBSMMHANLG-XZNUSECASA-N [[(2r,3s,4r,5r)-5-(6-aminopurin-9-yl)-3,4-dihydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl] [hydroxy-[1-(2-nitrophenyl)ethoxy]phosphoryl] hydrogen phosphate Chemical compound C([C@@H]1[C@H]([C@@H](O)[C@@H](O1)N1C2=NC=NC(N)=C2N=C1)O)OP(O)(=O)OP(O)(=O)OP(O)(=O)OC(C)C1=CC=CC=C1[N+]([O-])=O UOLVQBSMMHANLG-XZNUSECASA-N 0.000 description 6
- 230000002159 abnormal effect Effects 0.000 description 6
- 235000020958 biotin Nutrition 0.000 description 6
- 239000000356 contaminant Substances 0.000 description 6
- 230000001276 controlling effect Effects 0.000 description 6
- 230000000670 limiting effect Effects 0.000 description 6
- 235000019689 luncheon sausage Nutrition 0.000 description 6
- 244000052769 pathogen Species 0.000 description 6
- 102000004196 processed proteins & peptides Human genes 0.000 description 6
- 239000011535 reaction buffer Substances 0.000 description 6
- 239000003381 stabilizer Substances 0.000 description 6
- 101000749445 Enterobacteria phage T4 Recombination and repair protein Proteins 0.000 description 5
- CZPWVGJYEJSRLH-UHFFFAOYSA-N Pyrimidine Chemical compound C1=CN=CN=C1 CZPWVGJYEJSRLH-UHFFFAOYSA-N 0.000 description 5
- 101710183280 Topoisomerase Proteins 0.000 description 5
- 241000700605 Viruses Species 0.000 description 5
- 230000009286 beneficial effect Effects 0.000 description 5
- 210000004899 c-terminal region Anatomy 0.000 description 5
- 201000010099 disease Diseases 0.000 description 5
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 5
- 238000004108 freeze drying Methods 0.000 description 5
- 238000002955 isolation Methods 0.000 description 5
- 239000000463 material Substances 0.000 description 5
- 238000005457 optimization Methods 0.000 description 5
- 230000001717 pathogenic effect Effects 0.000 description 5
- 239000013612 plasmid Substances 0.000 description 5
- 229920000642 polymer Polymers 0.000 description 5
- 229920001184 polypeptide Polymers 0.000 description 5
- 150000003839 salts Chemical class 0.000 description 5
- 238000012163 sequencing technique Methods 0.000 description 5
- 230000002269 spontaneous effect Effects 0.000 description 5
- 230000003068 static effect Effects 0.000 description 5
- 229940113082 thymine Drugs 0.000 description 5
- 230000007704 transition Effects 0.000 description 5
- MIKUYHXYGGJMLM-GIMIYPNGSA-N Crotonoside Natural products C1=NC2=C(N)NC(=O)N=C2N1[C@H]1O[C@@H](CO)[C@H](O)[C@@H]1O MIKUYHXYGGJMLM-GIMIYPNGSA-N 0.000 description 4
- NYHBQMYGNKIUIF-UHFFFAOYSA-N D-guanosine Natural products C1=2NC(N)=NC(=O)C=2N=CN1C1OC(CO)C(O)C1O NYHBQMYGNKIUIF-UHFFFAOYSA-N 0.000 description 4
- 102220491783 High mobility group protein B1_K3A_mutation Human genes 0.000 description 4
- 108010093488 His-His-His-His-His-His Proteins 0.000 description 4
- PYMYPHUHKUWMLA-LMVFSUKVSA-N Ribose Natural products OC[C@@H](O)[C@@H](O)[C@@H](O)C=O PYMYPHUHKUWMLA-LMVFSUKVSA-N 0.000 description 4
- HMFHBZSHGGEWLO-UHFFFAOYSA-N alpha-D-Furanose-Ribose Natural products OCC1OC(O)C(O)C1O HMFHBZSHGGEWLO-UHFFFAOYSA-N 0.000 description 4
- 230000002238 attenuated effect Effects 0.000 description 4
- 230000000903 blocking effect Effects 0.000 description 4
- 210000004027 cell Anatomy 0.000 description 4
- 230000008859 change Effects 0.000 description 4
- 239000002299 complementary DNA Substances 0.000 description 4
- 230000001351 cycling effect Effects 0.000 description 4
- SUYVUBYJARFZHO-RRKCRQDMSA-N dATP Chemical compound C1=NC=2C(N)=NC=NC=2N1[C@H]1C[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 SUYVUBYJARFZHO-RRKCRQDMSA-N 0.000 description 4
- SUYVUBYJARFZHO-UHFFFAOYSA-N dATP Natural products C1=NC=2C(N)=NC=NC=2N1C1CC(O)C(COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 SUYVUBYJARFZHO-UHFFFAOYSA-N 0.000 description 4
- 238000009826 distribution Methods 0.000 description 4
- 230000007717 exclusion Effects 0.000 description 4
- 238000002866 fluorescence resonance energy transfer Methods 0.000 description 4
- 229940029575 guanosine Drugs 0.000 description 4
- 230000006872 improvement Effects 0.000 description 4
- 230000001939 inductive effect Effects 0.000 description 4
- 230000005764 inhibitory process Effects 0.000 description 4
- 230000010354 integration Effects 0.000 description 4
- 230000002085 persistent effect Effects 0.000 description 4
- 239000011295 pitch Substances 0.000 description 4
- 230000004044 response Effects 0.000 description 4
- 101150014817 ruvB gene Proteins 0.000 description 4
- 239000007787 solid Substances 0.000 description 4
- 238000010186 staining Methods 0.000 description 4
- 230000004936 stimulating effect Effects 0.000 description 4
- 230000000638 stimulation Effects 0.000 description 4
- 150000008163 sugars Chemical class 0.000 description 4
- 101710117642 Beta sliding clamp Proteins 0.000 description 3
- HMFHBZSHGGEWLO-SOOFDHNKSA-N D-ribofuranose Chemical compound OC[C@H]1OC(O)[C@H](O)[C@@H]1O HMFHBZSHGGEWLO-SOOFDHNKSA-N 0.000 description 3
- 230000033616 DNA repair Effects 0.000 description 3
- 238000001712 DNA sequencing Methods 0.000 description 3
- IAZDPXIOMUYVGZ-UHFFFAOYSA-N Dimethylsulphoxide Chemical compound CS(C)=O IAZDPXIOMUYVGZ-UHFFFAOYSA-N 0.000 description 3
- 101100300807 Drosophila melanogaster spn-A gene Proteins 0.000 description 3
- 101000621107 Enterobacteria phage N4 Probable portal protein Proteins 0.000 description 3
- 101000990000 Enterobacteria phage T4 Sliding clamp Proteins 0.000 description 3
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 3
- 108090000652 Flap endonucleases Proteins 0.000 description 3
- 102000004150 Flap endonucleases Human genes 0.000 description 3
- 102100034343 Integrase Human genes 0.000 description 3
- 108010092799 RNA-directed DNA polymerase Proteins 0.000 description 3
- 102000002933 Thioredoxin Human genes 0.000 description 3
- 230000001580 bacterial effect Effects 0.000 description 3
- 238000000576 coating method Methods 0.000 description 3
- NHVNXKFIZYSCEB-XLPZGREQSA-N dTTP Chemical compound O=C1NC(=O)C(C)=CN1[C@@H]1O[C@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)[C@@H](O)C1 NHVNXKFIZYSCEB-XLPZGREQSA-N 0.000 description 3
- 238000011161 development Methods 0.000 description 3
- 230000018109 developmental process Effects 0.000 description 3
- 238000002405 diagnostic procedure Methods 0.000 description 3
- 238000001962 electrophoresis Methods 0.000 description 3
- ZMMJGEGLRURXTF-UHFFFAOYSA-N ethidium bromide Chemical compound [Br-].C12=CC(N)=CC=C2C2=CC=C(N)C=C2[N+](CC)=C1C1=CC=CC=C1 ZMMJGEGLRURXTF-UHFFFAOYSA-N 0.000 description 3
- 229960005542 ethidium bromide Drugs 0.000 description 3
- 239000003112 inhibitor Substances 0.000 description 3
- 230000002401 inhibitory effect Effects 0.000 description 3
- 238000002372 labelling Methods 0.000 description 3
- 238000005259 measurement Methods 0.000 description 3
- 230000035515 penetration Effects 0.000 description 3
- 238000002360 preparation method Methods 0.000 description 3
- 238000010223 real-time analysis Methods 0.000 description 3
- 230000008439 repair process Effects 0.000 description 3
- 230000003252 repetitive effect Effects 0.000 description 3
- 238000011160 research Methods 0.000 description 3
- 238000010839 reverse transcription Methods 0.000 description 3
- 229920006395 saturated elastomer Polymers 0.000 description 3
- 238000009738 saturating Methods 0.000 description 3
- 238000012772 sequence design Methods 0.000 description 3
- 230000006641 stabilisation Effects 0.000 description 3
- 238000011105 stabilization Methods 0.000 description 3
- 238000005382 thermal cycling Methods 0.000 description 3
- 108060008226 thioredoxin Proteins 0.000 description 3
- 229940094937 thioredoxin Drugs 0.000 description 3
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 3
- HDTRYLNUVZCQOY-UHFFFAOYSA-N α-D-glucopyranosyl-α-D-glucopyranoside Natural products OC1C(O)C(O)C(CO)OC1OC1C(O)C(O)C(O)C(CO)O1 HDTRYLNUVZCQOY-UHFFFAOYSA-N 0.000 description 2
- QKNYBSVHEMOAJP-UHFFFAOYSA-N 2-amino-2-(hydroxymethyl)propane-1,3-diol;hydron;chloride Chemical compound Cl.OCC(N)(CO)CO QKNYBSVHEMOAJP-UHFFFAOYSA-N 0.000 description 2
- 108700028369 Alleles Proteins 0.000 description 2
- 241000193830 Bacillus <bacterium> Species 0.000 description 2
- 108050006400 Cyclin Proteins 0.000 description 2
- 108090000133 DNA helicases Proteins 0.000 description 2
- 102000003844 DNA helicases Human genes 0.000 description 2
- 101710135281 DNA polymerase III PolC-type Proteins 0.000 description 2
- 101710096438 DNA-binding protein Proteins 0.000 description 2
- AHCYMLUZIRLXAA-SHYZEUOFSA-N Deoxyuridine 5'-triphosphate Chemical compound O1[C@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)[C@@H](O)C[C@@H]1N1C(=O)NC(=O)C=C1 AHCYMLUZIRLXAA-SHYZEUOFSA-N 0.000 description 2
- 108010006296 DnaB Helicases Proteins 0.000 description 2
- 101000829157 Enterobacteria phage T4 Single-stranded DNA-binding protein Proteins 0.000 description 2
- 241000206602 Eukaryota Species 0.000 description 2
- 241000285023 Formosa Species 0.000 description 2
- 108010070675 Glutathione transferase Proteins 0.000 description 2
- 108010043121 Green Fluorescent Proteins Proteins 0.000 description 2
- 102000004144 Green Fluorescent Proteins Human genes 0.000 description 2
- 102100029100 Hematopoietic prostaglandin D synthase Human genes 0.000 description 2
- 101710175625 Maltose/maltodextrin-binding periplasmic protein Proteins 0.000 description 2
- 108010021466 Mutant Proteins Proteins 0.000 description 2
- 102000008300 Mutant Proteins Human genes 0.000 description 2
- 108091093037 Peptide nucleic acid Proteins 0.000 description 2
- ISWSIDIOOBJBQZ-UHFFFAOYSA-N Phenol Chemical compound OC1=CC=CC=C1 ISWSIDIOOBJBQZ-UHFFFAOYSA-N 0.000 description 2
- 102100036691 Proliferating cell nuclear antigen Human genes 0.000 description 2
- 241000193998 Streptococcus pneumoniae Species 0.000 description 2
- HDTRYLNUVZCQOY-WSWWMNSNSA-N Trehalose Natural products O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@@H]1O[C@@H]1[C@H](O)[C@@H](O)[C@@H](O)[C@@H](CO)O1 HDTRYLNUVZCQOY-WSWWMNSNSA-N 0.000 description 2
- 239000002253 acid Substances 0.000 description 2
- 230000002378 acidificating effect Effects 0.000 description 2
- 150000007513 acids Chemical class 0.000 description 2
- 230000002730 additional effect Effects 0.000 description 2
- 239000011543 agarose gel Substances 0.000 description 2
- HDTRYLNUVZCQOY-LIZSDCNHSA-N alpha,alpha-trehalose Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)O[C@@H]1O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 HDTRYLNUVZCQOY-LIZSDCNHSA-N 0.000 description 2
- 239000011324 bead Substances 0.000 description 2
- 108010005774 beta-Galactosidase Proteins 0.000 description 2
- 230000004071 biological effect Effects 0.000 description 2
- 210000004369 blood Anatomy 0.000 description 2
- 239000008280 blood Substances 0.000 description 2
- 239000007853 buffer solution Substances 0.000 description 2
- 239000008366 buffered solution Substances 0.000 description 2
- DSSYKIVIOFKYAU-UHFFFAOYSA-N camphor Chemical compound C1CC2(C)C(=O)CC1C2(C)C DSSYKIVIOFKYAU-UHFFFAOYSA-N 0.000 description 2
- 230000003197 catalytic effect Effects 0.000 description 2
- 210000000349 chromosome Anatomy 0.000 description 2
- 238000003776 cleavage reaction Methods 0.000 description 2
- 239000011248 coating agent Substances 0.000 description 2
- 239000005515 coenzyme Substances 0.000 description 2
- 238000001816 cooling Methods 0.000 description 2
- 239000008358 core component Substances 0.000 description 2
- 230000002596 correlated effect Effects 0.000 description 2
- CVSVTCORWBXHQV-UHFFFAOYSA-N creatine Chemical compound NC(=[NH2+])N(C)CC([O-])=O CVSVTCORWBXHQV-UHFFFAOYSA-N 0.000 description 2
- RGWHQCVHVJXOKC-SHYZEUOFSA-J dCTP(4-) Chemical compound O=C1N=C(N)C=CN1[C@@H]1O[C@H](COP([O-])(=O)OP([O-])(=O)OP([O-])([O-])=O)[C@@H](O)C1 RGWHQCVHVJXOKC-SHYZEUOFSA-J 0.000 description 2
- HAAZLUGHYHWQIW-KVQBGUIXSA-N dGTP Chemical compound C1=NC=2C(=O)NC(N)=NC=2N1[C@H]1C[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 HAAZLUGHYHWQIW-KVQBGUIXSA-N 0.000 description 2
- 230000003247 decreasing effect Effects 0.000 description 2
- 230000007812 deficiency Effects 0.000 description 2
- 230000000593 degrading effect Effects 0.000 description 2
- 230000001934 delay Effects 0.000 description 2
- 238000012217 deletion Methods 0.000 description 2
- 230000037430 deletion Effects 0.000 description 2
- 238000003936 denaturing gel electrophoresis Methods 0.000 description 2
- 230000001687 destabilization Effects 0.000 description 2
- 239000012502 diagnostic product Substances 0.000 description 2
- 238000009792 diffusion process Methods 0.000 description 2
- 229940079593 drug Drugs 0.000 description 2
- 239000003814 drug Substances 0.000 description 2
- 230000009977 dual effect Effects 0.000 description 2
- 230000005284 excitation Effects 0.000 description 2
- 238000000605 extraction Methods 0.000 description 2
- 230000002068 genetic effect Effects 0.000 description 2
- 238000003205 genotyping method Methods 0.000 description 2
- 239000001046 green dye Substances 0.000 description 2
- 239000005090 green fluorescent protein Substances 0.000 description 2
- 238000010438 heat treatment Methods 0.000 description 2
- 229920001519 homopolymer Polymers 0.000 description 2
- 230000002779 inactivation Effects 0.000 description 2
- 238000010348 incorporation Methods 0.000 description 2
- 238000013383 initial experiment Methods 0.000 description 2
- 238000012933 kinetic analysis Methods 0.000 description 2
- 125000001921 locked nucleotide group Chemical group 0.000 description 2
- 239000011777 magnesium Substances 0.000 description 2
- 230000004060 metabolic process Effects 0.000 description 2
- 244000005700 microbiome Species 0.000 description 2
- 244000045947 parasite Species 0.000 description 2
- 230000000737 periodic effect Effects 0.000 description 2
- 238000006303 photolysis reaction Methods 0.000 description 2
- 230000015843 photosynthesis, light reaction Effects 0.000 description 2
- 229920002401 polyacrylamide Polymers 0.000 description 2
- 230000000750 progressive effect Effects 0.000 description 2
- 230000004850 protein–protein interaction Effects 0.000 description 2
- 238000000746 purification Methods 0.000 description 2
- 238000011002 quantification Methods 0.000 description 2
- 238000004064 recycling Methods 0.000 description 2
- 238000005057 refrigeration Methods 0.000 description 2
- 230000007017 scission Effects 0.000 description 2
- 238000004904 shortening Methods 0.000 description 2
- 229940031000 streptococcus pneumoniae Drugs 0.000 description 2
- 230000002459 sustained effect Effects 0.000 description 2
- 230000001052 transient effect Effects 0.000 description 2
- 230000005945 translocation Effects 0.000 description 2
- NNMALANKTSRILL-LXENMSTPSA-N 3-[(2z,5e)-2-[[3-(2-carboxyethyl)-5-[(z)-[(3e,4r)-3-ethylidene-4-methyl-5-oxopyrrolidin-2-ylidene]methyl]-4-methyl-1h-pyrrol-2-yl]methylidene]-5-[(4-ethyl-3-methyl-5-oxopyrrol-2-yl)methylidene]-4-methylpyrrol-3-yl]propanoic acid Chemical compound O=C1C(CC)=C(C)C(\C=C\2C(=C(CCC(O)=O)C(=C/C3=C(C(C)=C(\C=C/4\C(\[C@@H](C)C(=O)N\4)=C\C)N3)CCC(O)=O)/N/2)C)=N1 NNMALANKTSRILL-LXENMSTPSA-N 0.000 description 1
- 101150072179 ATP1 gene Proteins 0.000 description 1
- 229920000936 Agarose Polymers 0.000 description 1
- 102000002260 Alkaline Phosphatase Human genes 0.000 description 1
- 108020004774 Alkaline Phosphatase Proteins 0.000 description 1
- 101710170230 Antimicrobial peptide 1 Proteins 0.000 description 1
- 241000207208 Aquifex Species 0.000 description 1
- 101100003366 Arabidopsis thaliana ATPA gene Proteins 0.000 description 1
- 108090001008 Avidin Proteins 0.000 description 1
- 101900090047 Bacillus subtilis DNA polymerase I Proteins 0.000 description 1
- 102100026189 Beta-galactosidase Human genes 0.000 description 1
- 108091003079 Bovine Serum Albumin Proteins 0.000 description 1
- 235000011299 Brassica oleracea var botrytis Nutrition 0.000 description 1
- 235000017647 Brassica oleracea var italica Nutrition 0.000 description 1
- 240000003259 Brassica oleracea var. botrytis Species 0.000 description 1
- 101000909256 Caldicellulosiruptor bescii (strain ATCC BAA-1888 / DSM 6725 / Z-1320) DNA polymerase I Proteins 0.000 description 1
- OKTJSMMVPCPJKN-UHFFFAOYSA-N Carbon Chemical group [C] OKTJSMMVPCPJKN-UHFFFAOYSA-N 0.000 description 1
- 229920002101 Chitin Polymers 0.000 description 1
- 241000606161 Chlamydia Species 0.000 description 1
- 108020004635 Complementary DNA Proteins 0.000 description 1
- 108020001019 DNA Primers Proteins 0.000 description 1
- 108020003215 DNA Probes Proteins 0.000 description 1
- 102000003915 DNA Topoisomerases Human genes 0.000 description 1
- 230000005778 DNA damage Effects 0.000 description 1
- 231100000277 DNA damage Toxicity 0.000 description 1
- 101710177611 DNA polymerase II large subunit Proteins 0.000 description 1
- 101710184669 DNA polymerase II small subunit Proteins 0.000 description 1
- 101710159156 DNA polymerase IV Proteins 0.000 description 1
- 239000003298 DNA probe Substances 0.000 description 1
- 230000004543 DNA replication Effects 0.000 description 1
- 102000004163 DNA-directed RNA polymerases Human genes 0.000 description 1
- 108090000626 DNA-directed RNA polymerases Proteins 0.000 description 1
- 229920002307 Dextran Polymers 0.000 description 1
- 102100021649 Elongator complex protein 6 Human genes 0.000 description 1
- 241000702224 Enterobacteria phage M13 Species 0.000 description 1
- 101000929572 Enterobacteria phage T4 ATP-dependent DNA helicase dda Proteins 0.000 description 1
- 241000709661 Enterovirus Species 0.000 description 1
- 241000702315 Escherichia virus phiX174 Species 0.000 description 1
- 241000186394 Eubacterium Species 0.000 description 1
- XZWYTXMRWQJBGX-VXBMVYAYSA-N FLAG peptide Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CCCCN)NC(=O)[C@@H](NC(=O)[C@@H](N)CC(O)=O)CC1=CC=C(O)C=C1 XZWYTXMRWQJBGX-VXBMVYAYSA-N 0.000 description 1
- 229920001917 Ficoll Polymers 0.000 description 1
- 241000233866 Fungi Species 0.000 description 1
- 101001057037 Gallus gallus Adenylate kinase isoenzyme 1 Proteins 0.000 description 1
- 206010071602 Genetic polymorphism Diseases 0.000 description 1
- HVLSXIKZNLPZJJ-TXZCQADKSA-N HA peptide Chemical compound C([C@@H](C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](C(C)C)C(=O)N1[C@@H](CCC1)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(=O)N[C@@H](C)C(O)=O)NC(=O)[C@H]1N(CCC1)C(=O)[C@@H](N)CC=1C=CC(O)=CC=1)C1=CC=C(O)C=C1 HVLSXIKZNLPZJJ-TXZCQADKSA-N 0.000 description 1
- 101710154606 Hemagglutinin Proteins 0.000 description 1
- 208000009889 Herpes Simplex Diseases 0.000 description 1
- 208000007514 Herpes zoster Diseases 0.000 description 1
- 101000889953 Homo sapiens Apolipoprotein B-100 Proteins 0.000 description 1
- 101100065219 Homo sapiens ELP6 gene Proteins 0.000 description 1
- 108010001336 Horseradish Peroxidase Proteins 0.000 description 1
- 241000725303 Human immunodeficiency virus Species 0.000 description 1
- 208000023105 Huntington disease Diseases 0.000 description 1
- 208000026350 Inborn Genetic disease Diseases 0.000 description 1
- 102000003960 Ligases Human genes 0.000 description 1
- 108090000364 Ligases Proteins 0.000 description 1
- 108090000856 Lyases Proteins 0.000 description 1
- 102000004317 Lyases Human genes 0.000 description 1
- 101100010166 Mus musculus Dok3 gene Proteins 0.000 description 1
- 102100038895 Myc proto-oncogene protein Human genes 0.000 description 1
- 101710135898 Myc proto-oncogene protein Proteins 0.000 description 1
- 241000187479 Mycobacterium tuberculosis Species 0.000 description 1
- 108020004711 Nucleic Acid Probes Proteins 0.000 description 1
- 241000243981 Onchocerca Species 0.000 description 1
- 108700020796 Oncogene Proteins 0.000 description 1
- 101710093908 Outer capsid protein VP4 Proteins 0.000 description 1
- 101710135467 Outer capsid protein sigma-1 Proteins 0.000 description 1
- 238000012408 PCR amplification Methods 0.000 description 1
- 238000002944 PCR assay Methods 0.000 description 1
- 241000287463 Phalacrocorax Species 0.000 description 1
- 108010010677 Phosphodiesterase I Proteins 0.000 description 1
- 102000001105 Phosphofructokinases Human genes 0.000 description 1
- 108010069341 Phosphofructokinases Proteins 0.000 description 1
- 108091000080 Phosphotransferase Proteins 0.000 description 1
- 241000224016 Plasmodium Species 0.000 description 1
- 208000000474 Poliomyelitis Diseases 0.000 description 1
- 239000004698 Polyethylene Substances 0.000 description 1
- 229920002560 Polyethylene Glycol 3000 Polymers 0.000 description 1
- 239000004372 Polyvinyl alcohol Substances 0.000 description 1
- 208000032236 Predisposition to disease Diseases 0.000 description 1
- 101710176177 Protein A56 Proteins 0.000 description 1
- 101800004937 Protein C Proteins 0.000 description 1
- 102000002067 Protein Subunits Human genes 0.000 description 1
- 108010001267 Protein Subunits Proteins 0.000 description 1
- 241000588770 Proteus mirabilis Species 0.000 description 1
- 241000589517 Pseudomonas aeruginosa Species 0.000 description 1
- 241000205223 Pyrobaculum islandicum Species 0.000 description 1
- 101000902592 Pyrococcus furiosus (strain ATCC 43587 / DSM 3638 / JCM 8422 / Vc1) DNA polymerase Proteins 0.000 description 1
- 238000011529 RT qPCR Methods 0.000 description 1
- 206010037742 Rabies Diseases 0.000 description 1
- 108700018273 Rad30 Proteins 0.000 description 1
- 108091036333 Rapid DNA Proteins 0.000 description 1
- 102000007056 Recombinant Fusion Proteins Human genes 0.000 description 1
- 108010008281 Recombinant Fusion Proteins Proteins 0.000 description 1
- 108091081062 Repeated sequence (DNA) Proteins 0.000 description 1
- 102000006382 Ribonucleases Human genes 0.000 description 1
- 108010083644 Ribonucleases Proteins 0.000 description 1
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 1
- 235000014680 Saccharomyces cerevisiae Nutrition 0.000 description 1
- 102100036546 Salivary acidic proline-rich phosphoprotein 1/2 Human genes 0.000 description 1
- 241000607142 Salmonella Species 0.000 description 1
- 241000293869 Salmonella enterica subsp. enterica serovar Typhimurium Species 0.000 description 1
- 101800001700 Saposin-D Proteins 0.000 description 1
- BQCADISMDOOEFD-UHFFFAOYSA-N Silver Chemical compound [Ag] BQCADISMDOOEFD-UHFFFAOYSA-N 0.000 description 1
- 241000589196 Sinorhizobium meliloti Species 0.000 description 1
- 101710119335 Sliding-clamp-loader large subunit Proteins 0.000 description 1
- 101710087759 Sliding-clamp-loader small subunit Proteins 0.000 description 1
- 108010090804 Streptavidin Proteins 0.000 description 1
- 241000194017 Streptococcus Species 0.000 description 1
- 229930006000 Sucrose Natural products 0.000 description 1
- CZMRCDWAGMRECN-UGDNZRGBSA-N Sucrose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 CZMRCDWAGMRECN-UGDNZRGBSA-N 0.000 description 1
- 101710194518 T4 protein Proteins 0.000 description 1
- 241001235254 Thermococcus kodakarensis Species 0.000 description 1
- 241000204666 Thermotoga maritima Species 0.000 description 1
- 241000589500 Thermus aquaticus Species 0.000 description 1
- 241000589499 Thermus thermophilus Species 0.000 description 1
- IQFYYKKMVGJFEH-XLPZGREQSA-N Thymidine Chemical group O=C1NC(=O)C(C)=CN1[C@@H]1O[C@H](CO)[C@@H](O)C1 IQFYYKKMVGJFEH-XLPZGREQSA-N 0.000 description 1
- 108010001244 Tli polymerase Proteins 0.000 description 1
- 241000223997 Toxoplasma gondii Species 0.000 description 1
- 101710150448 Transcriptional regulator Myc Proteins 0.000 description 1
- 239000007983 Tris buffer Substances 0.000 description 1
- 241000223104 Trypanosoma Species 0.000 description 1
- 108700025716 Tumor Suppressor Genes Proteins 0.000 description 1
- 102000044209 Tumor Suppressor Genes Human genes 0.000 description 1
- PGAVKCOVUIYSFO-XVFCMESISA-N UTP Chemical compound O[C@@H]1[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O[C@H]1N1C(=O)NC(=O)C=C1 PGAVKCOVUIYSFO-XVFCMESISA-N 0.000 description 1
- 244000301083 Ustilago maydis Species 0.000 description 1
- 235000015919 Ustilago maydis Nutrition 0.000 description 1
- 206010046865 Vaccinia virus infection Diseases 0.000 description 1
- 241000700647 Variola virus Species 0.000 description 1
- 241000251539 Vertebrata <Metazoa> Species 0.000 description 1
- 241001376713 Xenops Species 0.000 description 1
- HCIKUKNAJRJFOW-KQYNXXCUSA-N [(2r,3s,4r,5r)-5-(6-aminopurin-9-yl)-3,4-dihydroxyoxolan-2-yl]methyl dihydroxyphosphinothioyl hydrogen phosphate Chemical compound C1=NC=2C(N)=NC=NC=2N1[C@@H]1O[C@H](COP(O)(=O)OP(O)(O)=S)[C@@H](O)[C@H]1O HCIKUKNAJRJFOW-KQYNXXCUSA-N 0.000 description 1
- HDRRAMINWIWTNU-NTSWFWBYSA-N [[(2s,5r)-5-(2-amino-6-oxo-3h-purin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl] phosphono hydrogen phosphate Chemical compound C1=2NC(N)=NC(=O)C=2N=CN1[C@H]1CC[C@@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 HDRRAMINWIWTNU-NTSWFWBYSA-N 0.000 description 1
- ARLKCWCREKRROD-POYBYMJQSA-N [[(2s,5r)-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl] phosphono hydrogen phosphate Chemical compound O=C1N=C(N)C=CN1[C@@H]1O[C@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)CC1 ARLKCWCREKRROD-POYBYMJQSA-N 0.000 description 1
- 238000003916 acid precipitation Methods 0.000 description 1
- 239000013543 active substance Substances 0.000 description 1
- 230000002411 adverse Effects 0.000 description 1
- 125000003295 alanine group Chemical group N[C@@H](C)C(=O)* 0.000 description 1
- 125000003158 alcohol group Chemical group 0.000 description 1
- 230000001195 anabolic effect Effects 0.000 description 1
- 230000003712 anti-aging effect Effects 0.000 description 1
- 239000000427 antigen Substances 0.000 description 1
- 102000036639 antigens Human genes 0.000 description 1
- 108091007433 antigens Proteins 0.000 description 1
- QVGXLLKOCUKJST-UHFFFAOYSA-N atomic oxygen Chemical group [O] QVGXLLKOCUKJST-UHFFFAOYSA-N 0.000 description 1
- 101150105046 atpI gene Proteins 0.000 description 1
- 238000013475 authorization Methods 0.000 description 1
- 230000004888 barrier function Effects 0.000 description 1
- WQZGKKKJIJFFOK-FPRJBGLDSA-N beta-D-galactose Chemical compound OC[C@H]1O[C@@H](O)[C@H](O)[C@@H](O)[C@H]1O WQZGKKKJIJFFOK-FPRJBGLDSA-N 0.000 description 1
- 238000012742 biochemical analysis Methods 0.000 description 1
- 230000031018 biological processes and functions Effects 0.000 description 1
- 230000033228 biological regulation Effects 0.000 description 1
- 239000012472 biological sample Substances 0.000 description 1
- 230000036760 body temperature Effects 0.000 description 1
- 229940098773 bovine serum albumin Drugs 0.000 description 1
- 239000006227 byproduct Substances 0.000 description 1
- 238000010804 cDNA synthesis Methods 0.000 description 1
- 229910052799 carbon Inorganic materials 0.000 description 1
- 238000004587 chromatography analysis Methods 0.000 description 1
- 238000004440 column chromatography Methods 0.000 description 1
- 239000012141 concentrate Substances 0.000 description 1
- 239000012468 concentrated sample Substances 0.000 description 1
- 239000004020 conductor Substances 0.000 description 1
- 238000012790 confirmation Methods 0.000 description 1
- 238000010276 construction Methods 0.000 description 1
- 238000010924 continuous production Methods 0.000 description 1
- 239000013068 control sample Substances 0.000 description 1
- 230000009146 cooperative binding Effects 0.000 description 1
- 230000002079 cooperative effect Effects 0.000 description 1
- 230000000875 corresponding effect Effects 0.000 description 1
- 229960003624 creatine Drugs 0.000 description 1
- 239000006046 creatine Substances 0.000 description 1
- 125000004122 cyclic group Chemical group 0.000 description 1
- 230000006378 damage Effects 0.000 description 1
- URGJWIFLBWJRMF-JGVFFNPUSA-N ddTTP Chemical compound O=C1NC(=O)C(C)=CN1[C@@H]1O[C@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)CC1 URGJWIFLBWJRMF-JGVFFNPUSA-N 0.000 description 1
- 238000000354 decomposition reaction Methods 0.000 description 1
- 230000002950 deficient Effects 0.000 description 1
- 230000002939 deleterious effect Effects 0.000 description 1
- 238000004925 denaturation Methods 0.000 description 1
- 230000036425 denaturation Effects 0.000 description 1
- 230000008021 deposition Effects 0.000 description 1
- 230000000368 destabilizing effect Effects 0.000 description 1
- 238000003745 diagnosis Methods 0.000 description 1
- 230000029087 digestion Effects 0.000 description 1
- 150000002016 disaccharides Chemical class 0.000 description 1
- 239000011363 dried mixture Substances 0.000 description 1
- 230000008846 dynamic interplay Effects 0.000 description 1
- 238000001493 electron microscopy Methods 0.000 description 1
- 230000007613 environmental effect Effects 0.000 description 1
- 238000001976 enzyme digestion Methods 0.000 description 1
- 238000011156 evaluation Methods 0.000 description 1
- 230000029142 excretion Effects 0.000 description 1
- 230000002349 favourable effect Effects 0.000 description 1
- 239000012467 final product Substances 0.000 description 1
- GNBHRKFJIUUOQI-UHFFFAOYSA-N fluorescein Chemical compound O1C(=O)C2=CC=CC=C2C21C1=CC=C(O)C=C1OC1=CC(O)=CC=C21 GNBHRKFJIUUOQI-UHFFFAOYSA-N 0.000 description 1
- 238000000198 fluorescence anisotropy Methods 0.000 description 1
- 238000005194 fractionation Methods 0.000 description 1
- 231100000221 frame shift mutation induction Toxicity 0.000 description 1
- 230000037433 frameshift Effects 0.000 description 1
- 102000037865 fusion proteins Human genes 0.000 description 1
- 108020001507 fusion proteins Proteins 0.000 description 1
- 230000014509 gene expression Effects 0.000 description 1
- 230000030279 gene silencing Effects 0.000 description 1
- 238000007429 general method Methods 0.000 description 1
- 230000008570 general process Effects 0.000 description 1
- 238000012252 genetic analysis Methods 0.000 description 1
- 208000016361 genetic disease Diseases 0.000 description 1
- 239000011521 glass Substances 0.000 description 1
- 239000000185 hemagglutinin Substances 0.000 description 1
- 208000006454 hepatitis Diseases 0.000 description 1
- 231100000283 hepatitis Toxicity 0.000 description 1
- 208000005252 hepatitis A Diseases 0.000 description 1
- 208000002672 hepatitis B Diseases 0.000 description 1
- 238000004128 high performance liquid chromatography Methods 0.000 description 1
- 238000012203 high throughput assay Methods 0.000 description 1
- HNDVDQJCIGZPNO-UHFFFAOYSA-N histidine Natural products OC(=O)C(N)CC1=CN=CN1 HNDVDQJCIGZPNO-UHFFFAOYSA-N 0.000 description 1
- 102000052249 human APOB Human genes 0.000 description 1
- 230000001771 impaired effect Effects 0.000 description 1
- 238000011065 in-situ storage Methods 0.000 description 1
- 206010022000 influenza Diseases 0.000 description 1
- 238000003780 insertion Methods 0.000 description 1
- 230000037431 insertion Effects 0.000 description 1
- 230000009878 intermolecular interaction Effects 0.000 description 1
- 230000008863 intramolecular interaction Effects 0.000 description 1
- 150000002500 ions Chemical class 0.000 description 1
- 239000003446 ligand Substances 0.000 description 1
- 239000012035 limiting reagent Substances 0.000 description 1
- 125000005647 linker group Chemical group 0.000 description 1
- 239000007788 liquid Substances 0.000 description 1
- 239000007791 liquid phase Substances 0.000 description 1
- 230000005923 long-lasting effect Effects 0.000 description 1
- 239000006166 lysate Substances 0.000 description 1
- 229910001425 magnesium ion Inorganic materials 0.000 description 1
- 238000004949 mass spectrometry Methods 0.000 description 1
- 239000011159 matrix material Substances 0.000 description 1
- 239000012528 membrane Substances 0.000 description 1
- 238000002493 microarray Methods 0.000 description 1
- 230000000813 microbial effect Effects 0.000 description 1
- 238000002156 mixing Methods 0.000 description 1
- 238000001823 molecular biology technique Methods 0.000 description 1
- 238000010369 molecular cloning Methods 0.000 description 1
- 230000004001 molecular interaction Effects 0.000 description 1
- 239000003068 molecular probe Substances 0.000 description 1
- 125000004573 morpholin-4-yl group Chemical group N1(CCOCC1)* 0.000 description 1
- 229930027945 nicotinamide-adenine dinucleotide Natural products 0.000 description 1
- BOPGDPNILDQYTO-NNYOXOHSSA-N nicotinamide-adenine dinucleotide Chemical compound C1=CCC(C(=O)N)=CN1[C@H]1[C@H](O)[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OC[C@@H]2[C@H]([C@@H](O)[C@@H](O2)N2C3=NC=NC(N)=C3N=C2)O)O1 BOPGDPNILDQYTO-NNYOXOHSSA-N 0.000 description 1
- 239000002853 nucleic acid probe Substances 0.000 description 1
- 238000001821 nucleic acid purification Methods 0.000 description 1
- 238000005580 one pot reaction Methods 0.000 description 1
- 230000003287 optical effect Effects 0.000 description 1
- 238000012803 optimization experiment Methods 0.000 description 1
- 230000002018 overexpression Effects 0.000 description 1
- 229910052760 oxygen Inorganic materials 0.000 description 1
- 239000001301 oxygen Substances 0.000 description 1
- LCCNCVORNKJIRZ-UHFFFAOYSA-N parathion Chemical compound CCOP(=S)(OCC)OC1=CC=C([N+]([O-])=O)C=C1 LCCNCVORNKJIRZ-UHFFFAOYSA-N 0.000 description 1
- 230000036961 partial effect Effects 0.000 description 1
- 230000037361 pathway Effects 0.000 description 1
- 230000002688 persistence Effects 0.000 description 1
- 238000005191 phase separation Methods 0.000 description 1
- 125000002467 phosphate group Chemical group [H]OP(=O)(O[H])O[*] 0.000 description 1
- PTMHPRAIXMAOOB-UHFFFAOYSA-L phosphoramidate Chemical compound NP([O-])([O-])=O PTMHPRAIXMAOOB-UHFFFAOYSA-L 0.000 description 1
- 150000003013 phosphoric acid derivatives Chemical group 0.000 description 1
- 102000020233 phosphotransferase Human genes 0.000 description 1
- 230000003711 photoprotective effect Effects 0.000 description 1
- 230000000704 physical effect Effects 0.000 description 1
- 230000010399 physical interaction Effects 0.000 description 1
- 229920003023 plastic Polymers 0.000 description 1
- 239000004033 plastic Substances 0.000 description 1
- 239000002574 poison Substances 0.000 description 1
- 231100000614 poison Toxicity 0.000 description 1
- 229920002451 polyvinyl alcohol Polymers 0.000 description 1
- 239000011148 porous material Substances 0.000 description 1
- 238000001556 precipitation Methods 0.000 description 1
- 239000002987 primer (paints) Substances 0.000 description 1
- 239000003223 protective agent Substances 0.000 description 1
- 230000004952 protein activity Effects 0.000 description 1
- 229960000856 protein c Drugs 0.000 description 1
- 230000004853 protein function Effects 0.000 description 1
- 150000003230 pyrimidines Chemical class 0.000 description 1
- 238000012175 pyrosequencing Methods 0.000 description 1
- 238000004445 quantitative analysis Methods 0.000 description 1
- 238000010791 quenching Methods 0.000 description 1
- 230000000171 quenching effect Effects 0.000 description 1
- 238000011897 real-time detection Methods 0.000 description 1
- 101150002589 recF gene Proteins 0.000 description 1
- 101150066870 recG gene Proteins 0.000 description 1
- 102000005962 receptors Human genes 0.000 description 1
- 230000001172 regenerating effect Effects 0.000 description 1
- 230000001105 regulatory effect Effects 0.000 description 1
- 230000003362 replicative effect Effects 0.000 description 1
- PYWVYCXTNDRMGF-UHFFFAOYSA-N rhodamine B Chemical compound [Cl-].C=12C=CC(=[N+](CC)CC)C=C2OC2=CC(N(CC)CC)=CC=C2C=1C1=CC=CC=C1C(O)=O PYWVYCXTNDRMGF-UHFFFAOYSA-N 0.000 description 1
- 125000000548 ribosyl group Chemical group C1([C@H](O)[C@H](O)[C@H](O1)CO)* 0.000 description 1
- 230000005070 ripening Effects 0.000 description 1
- 201000005404 rubella Diseases 0.000 description 1
- 101150061752 ruvA gene Proteins 0.000 description 1
- 238000007480 sanger sequencing Methods 0.000 description 1
- 238000011896 sensitive detection Methods 0.000 description 1
- 238000007086 side reaction Methods 0.000 description 1
- 229910052709 silver Inorganic materials 0.000 description 1
- 239000004332 silver Substances 0.000 description 1
- 238000001542 size-exclusion chromatography Methods 0.000 description 1
- 230000011273 social behavior Effects 0.000 description 1
- 239000002904 solvent Substances 0.000 description 1
- 238000000638 solvent extraction Methods 0.000 description 1
- 108010068698 spleen exonuclease Proteins 0.000 description 1
- 230000028070 sporulation Effects 0.000 description 1
- 238000010561 standard procedure Methods 0.000 description 1
- 239000007858 starting material Substances 0.000 description 1
- 238000003860 storage Methods 0.000 description 1
- 239000005720 sucrose Substances 0.000 description 1
- 230000008093 supporting effect Effects 0.000 description 1
- 230000001629 suppression Effects 0.000 description 1
- 230000001360 synchronised effect Effects 0.000 description 1
- 230000002195 synergetic effect Effects 0.000 description 1
- 230000002194 synthesizing effect Effects 0.000 description 1
- 230000002123 temporal effect Effects 0.000 description 1
- 125000003396 thiol group Chemical group [H]S* 0.000 description 1
- UNXRWKVEANCORM-UHFFFAOYSA-N triphosphoric acid Chemical compound OP(O)(=O)OP(O)(=O)OP(O)(O)=O UNXRWKVEANCORM-UHFFFAOYSA-N 0.000 description 1
- LENZDBCJOHFCAS-UHFFFAOYSA-N tris Chemical compound OCC(N)(CO)CO LENZDBCJOHFCAS-UHFFFAOYSA-N 0.000 description 1
- 230000007306 turnover Effects 0.000 description 1
- 241001515965 unidentified phage Species 0.000 description 1
- 238000011144 upstream manufacturing Methods 0.000 description 1
- 208000007089 vaccinia Diseases 0.000 description 1
Images
Classifications
-
- B—PERFORMING OPERATIONS; TRANSPORTING
- B82—NANOTECHNOLOGY
- B82Y—SPECIFIC USES OR APPLICATIONS OF NANOSTRUCTURES; MEASUREMENT OR ANALYSIS OF NANOSTRUCTURES; MANUFACTURE OR TREATMENT OF NANOSTRUCTURES
- B82Y5/00—Nanobiotechnology or nanomedicine, e.g. protein engineering or drug delivery
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6813—Hybridisation assays
- C12Q1/6827—Hybridisation assays for detection of mutation or polymorphism
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6844—Nucleic acid amplification reactions
-
- B—PERFORMING OPERATIONS; TRANSPORTING
- B01—PHYSICAL OR CHEMICAL PROCESSES OR APPARATUS IN GENERAL
- B01L—CHEMICAL OR PHYSICAL LABORATORY APPARATUS FOR GENERAL USE
- B01L3/00—Containers or dishes for laboratory use, e.g. laboratory glassware; Droppers
- B01L3/50—Containers for the purpose of retaining a material to be analysed, e.g. test tubes
- B01L3/502—Containers for the purpose of retaining a material to be analysed, e.g. test tubes with fluid transport, e.g. in multi-compartment structures
- B01L3/5023—Containers for the purpose of retaining a material to be analysed, e.g. test tubes with fluid transport, e.g. in multi-compartment structures with a sample being transported to, and subsequently stored in an absorbent for analysis
-
- B—PERFORMING OPERATIONS; TRANSPORTING
- B01—PHYSICAL OR CHEMICAL PROCESSES OR APPARATUS IN GENERAL
- B01L—CHEMICAL OR PHYSICAL LABORATORY APPARATUS FOR GENERAL USE
- B01L7/00—Heating or cooling apparatus; Heat insulating devices
-
- B—PERFORMING OPERATIONS; TRANSPORTING
- B82—NANOTECHNOLOGY
- B82Y—SPECIFIC USES OR APPLICATIONS OF NANOSTRUCTURES; MEASUREMENT OR ANALYSIS OF NANOSTRUCTURES; MANUFACTURE OR TREATMENT OF NANOSTRUCTURES
- B82Y10/00—Nanotechnology for information processing, storage or transmission, e.g. quantum computing or single electron logic
Landscapes
- Chemical & Material Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Engineering & Computer Science (AREA)
- Organic Chemistry (AREA)
- Health & Medical Sciences (AREA)
- Wood Science & Technology (AREA)
- Zoology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- General Engineering & Computer Science (AREA)
- Biotechnology (AREA)
- Biophysics (AREA)
- Molecular Biology (AREA)
- General Health & Medical Sciences (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Microbiology (AREA)
- Physics & Mathematics (AREA)
- Analytical Chemistry (AREA)
- Biochemistry (AREA)
- Genetics & Genomics (AREA)
- Immunology (AREA)
- Nanotechnology (AREA)
- Crystallography & Structural Chemistry (AREA)
- Medicinal Chemistry (AREA)
- Pharmacology & Pharmacy (AREA)
- Medical Informatics (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Measuring Or Testing Involving Enzymes Or Micro-Organisms (AREA)
- Apparatus Associated With Microorganisms And Enzymes (AREA)
- Enzymes And Modification Thereof (AREA)
- Investigating, Analyzing Materials By Fluorescence Or Luminescence (AREA)
- Investigating Or Analysing Materials By The Use Of Chemical Reactions (AREA)
Description
DNAの増幅能力は、現代の生物学的および医学的研究の中心にある。これは、ほとんどの分子生物学手法が、アッセイの感度を増大するために、またはさらなる処理に充分な材料を準備するために多量の同一分子を含有する試料に依存するためである。種々の核酸増幅手法のうち、ポリメラーゼ連鎖反応(PCR)は、その感度と効率のため、短い核酸配列の増幅において最もよく用いられている。
このようなRecA/dsDNA複合体は、続いて侵入および合成のラウンドを開始するのに用いるRecA/ssDNAプライマー複合体によって標的化される正確な部位である。実際、RecAが結合した状態では、中間体はポリメラーゼに接近し得ず、dsDNAは、RecA/ssDNAプライマー複合体によって確実にはそれ以上侵入され得なくなり、したがって、この点から増幅可能でなくなる。さらに、このような鋳型からの合成は、RecAのない鋳型の他方の末端開始された場合に起こり得、これは、最終的に、結合RecAの物理的置換をもたらし得る。しかしながら、多くのポリメラーゼがこのような様式でRecAを置換し得るのか否かは明らかでない。またさらに、その合成ラウンドの開始部位は、代わりに今度は「ブロックされる」。かかる状況では、増幅は、時間に対して線形であるにすぎず、主に単鎖DNA増幅産物を生成する。
本発明は、RPAと称するDNAの増幅方法を提供し、以下の工程を含む。第1に、リコンビナーゼ因子を、第1および第2の核酸プライマーと接触させ、第1および第2の核タンパク質プライマーを形成する。第2に、第1および第2の核タンパク質プライマーを二本鎖標的配列に接触させて、前記第1の鎖の第1の部分に第1の二本鎖構造を形成させ、そして前記第2の鎖の第2の部分に二本鎖構造を形成させ、前記第1の核酸プライマーおよび前記第2の核酸プライマーの3’末端が、所与の鋳型DNA分子で互いに向かって配向される。第3に、前記第1および第2の核タンパク質プライマーの3’末端はDNAポリメラーゼによって伸長され、第1および第2の二本鎖核酸ならびに核酸の第1および第2の被置換鎖を生成させる。最後に、第2および第3の工程を所望の増幅度に達するまで反復する。
本発明は、標的核酸ポリマーの増幅のための方法の1つであるリコンビナーゼ−ポリメラーゼ増幅(RPA)を提供する。また、本発明は、高いリコンビナーゼ活性が高度に動的な組換え環境において維持されており、ATPによって補助される一般的なインビトロ環境を提供する。RPAの利点の1つは、二本鎖鋳型の熱融解を必要とせずに行い得ることである。したがって、高価なサーモサイクラーの必要性も排除される。本発明では、RPAが標的核酸ポリマーの指数関数的増幅を可能にするように構成され得る2つの関連するストラテジーを記載する。
リーディング鎖リコンビナーゼ−ポリメラーゼ増幅(lsRPA)では、単鎖または部分的に単鎖の核酸プライマーを、相同な二本鎖または部分的に二本鎖の配列にリコンビナーゼ因子を用いて標的化させ、これによりDループ構造が形成され得る。Dループの一部である侵入性単鎖プライマーを用い、ポリメラーゼ合成反応を開始させる。単一のプライマー種が標的核酸配列を、多数回の二本鎖侵入の後、合成を行なうことにより増幅する。2種類の対向するプライマーが用いられる場合、断片(標的配列)の増幅が達成され得る。LsRPAを、簡単に図1および2に示す。
1.[非標的核酸]−[標的核酸] −[非標的核酸]
2.[非標的核酸]−[標的核酸]
3.[標的核酸]−[非標的核酸]
4.[標的核酸]
のいずれかであり得る。
RPAは、RecAまたは機能的ホモログ(例えば、T4 uvsXタンパク質など)で被覆された合成オリゴヌクレオチドを用いた配列標的化により開始される。指数関数的増幅を可能にするためには、2つのかかる合成オリゴヌクレオチドは、それらの遊離3’末端が互いに向かって配向されるように使用し得る。このようなオリゴヌクレオチドおよびリコンビナーゼタンパク質を含む核タンパク質フィラメントは、複合体DNA内の標的を、迅速かつ特異的に同定する。いったん標的化されると、リコンビナーゼタンパク質は鎖交換を触媒し、その結果、Dループ構造が形成される。効率的な増幅のための手順には、ATPγSではなくATPを用いることが必要であり得る。ATPを用いる場合、RecO、RecR,および/またはRecFの分子が効率的な増幅に不可欠であることが示され得、またはuvsXリコンビナーゼを用いる場合は、uvsYタンパク質が使用され得る。
DNAポリメラーゼは、侵入性オリゴヌクレオチドと鋳型DNA間のハイブリッドを検出し、これに結合し、ハイブリッドにおいて露出された遊離3’−ヒドロキシルからのDNA合成を開始する。この3’−ヒドロキシルの露出および続くDNA合成は、鎖交換によって形成された二本鎖ハイブリッドからのリコンビナーゼタンパク質の分解を必要とすることがあり得る。この分解が達成されるためには、おそらく、侵入複合体からのリコンビナーゼの自発的分解を補助し得るATPを用いることが必要であり得る。さらに、分解は、反応混合物中に含める他のタンパク質(例えば、RuvA、RuvB、RuvC、recG、他のヘリカーゼ)または他の刺激成分(これらは、リコンビナーゼを鎖交換産物から外す機能を果たし得る)の使用によって刺激/増強され得る。
DNAポリメラーゼが、侵入性オリゴヌクレオチド、またはそれらの部分的に伸長された産物の遊離3’−ヒドロキシルを用いて鋳型DNAの相補的コピー合成すると、該ポリメラーゼは、反応物中に含まれた単一の鎖結合タンパク質(SSB)で被覆されたものであり得る単鎖DNAと置換される。理想的な構成では、標的核酸配列の両末端でのオリゴヌクレオチドの侵入が同様の時間枠内で起こり、その結果、同じ鋳型核酸上の2つのポリメラーゼがまず互いに向かって進行する。これらの伸長している複合体が互いに出合うと、元の鋳型は簡単に離れ落ち、ポリメラーゼは、鎖置換の必要なく合成を継続し、今度は、SSB結合ssDNA鋳型をコピーする。立体障害のため、両ポリメラーゼが出合うと、ポリメラーゼは一時的に鋳型から分離された状態となり得、2つの鋳型鎖の分離が可能となる。
いったん鋳型鎖が離れると、ポリメラーゼは、鋳型の末端までの伸長を終了し得る(または、最初の鋳型が所望の産物より長い場合は、第2の対向標的化部位としての機能を果たす配列を通過する)。指数関数的増幅を可能にするためには、新たな産物が、元の鋳型と同様に、両方の標的化末端からの様式で標的化および複製されることが必要である。新たに合成された標的化部位は、標的化リコンビナーゼ/オリゴヌクレオチドフィラメントに自由に利用可能である。また、合成のプライミングに最初に使用された部位は、反応において、鎖交換産物からのリコンビナーゼの分解に有利な条件の使用の結果として遊離状態のはずである。この後者の部位での再侵入は、該ポリメラーゼが第2の標的化部位を通過して合成され、この第2の部位でプライミングされ、そして第1の部位に戻るのに要した時間より短い時間で起こると、単鎖DNAが主要産物でなくなり、指数関数的増幅が起こる。多数の合成複合体が同じ鋳型上で機能すると、非常に短い増幅時間が達成され得る可能性が生じる。
(リーディング鎖RPA)lsRPAの本発明者らの記載において、本発明者らは、標的化配列を再生し、したがって、二本鎖DNAの指数関数的増幅を可能にする能力を有する多成分系について詳細に示す。Zarling法とは異なり、lsRPAは、単鎖DNAの線状産生を回避する。この問題を解決するため、単鎖産物の可能性および同時末端開始の必要性を完全に回避する別のアプローチがある。この方法は、必然的に、より複合的な反応混合物を伴う。それにもかかわらず、この場合、必要とされる成分はすべて、充分理解されており、単一の系に合成されやすいはずである。この系は、細胞の正常な複製サイクル中に起こる事象を反復し、リーディング鎖/ラギング鎖連結型合成を可能にする。この方法リーディング鎖/ラギング鎖RPAを、簡単に図1および3に示す。
RPAは、RecAまたはT4 uvsXまたは機能的ホモログで被覆された合成オリゴヌクレオチドを用いた配列標的化により開始される。かかる核タンパク質フィラメントは、複合体DNA内の標的を、迅速かつ特異的に同定する。いったん標的化されると、RecAまたはuvsXタンパク質は鎖交換を触媒し、その結果、Dループ構造が形成される。効率的な増幅のための手順には、ATPγSではなくATPを用いることが必要であり得る。しかしながら、リーディング鎖合成とラギング鎖合成の連携は、合成開始後に非常に迅速にリコンビナーゼが外されることの必要性を不要にし得る。ATPを用いる場合、RecO、RecR、およびRecFを細菌のrecAリコンビナーゼとともに用いる必要があり得、またはT4 uvsYタンパク質が、T4 uvsXタンパク質の場合の効率的な増幅に不可欠であることが示され得る。
プライモソームはDループで合成され得る。通常、大腸菌では、Dループ構造はRecAによって、インビボでの損傷DNAを救済する機構の一部として、または他の形態の組換え中に形成される。RecA媒介性鎖交換およびプライモソーム合成の組合せ作用の目的は、複製フォークを生成させることである。複製フォークは、分離された鋳型DNA鎖およびレプリソームを含む核タンパク質構造である。レプリソームは、ポリメラーゼホロ酵素複合体、プライモソーム、および両方の鋳型DNA鎖を同時に複製するのに必要とされる他の成分からなる。プライモソームは、複製フォーク進行に必要なDNA巻き解きおよびオカザキ断片プライミング機能の両方を提供する。同様のプライモソーム合成が、gp59およびgp41タンパク質によって指向されるT4ファージの組換え中間体において起こる。
複製フォークは、プライモソーム合成部位で合成される。大腸菌では、Dループの侵入鎖上の遊離3’末端の存在により、先に記載のDnaXクランプ負荷体複合体が刺激され、β−二量体がこの部位に合成され、スライディングクランプとして作用する。ホロ酵素および2つのコア単位は、骨格τサブユニットによって一緒に連結されている。τサブユニットはまた、β−二量体、クランプ負荷体、およびプライモソームのDnaBヘリカーゼ成分のための相互作用表面を有する。このような多重相互作用は、2つの非対称に連結されたコアポリメラーゼ複合体を用いてリーディング鎖およびラギング鎖の両方の合成を調和させるのに必要である。uvsYおよびgp32タンパク質ならびに他の成分を伴うT4ファージgp59/41タンパク質では、gp44およびgp62タンパク質によって助長されるスライディングクランプgp45の調和合成によってレプリソーム合成が開始される。
RPAでは、複製は2つの遠隔部位で開始され、複製フォークは、互いに向かって配向される。複製フォークが1点に集中すると、2つの元の鋳型鎖は、各フォークの後方および前方の両方において完全に分離された状態となり、互いから解離する。次いで、各フォークのリーディング鎖コアは合成を終了し、残存するRNAプライマーがプロセッシングされ、最終生成物が2つの二本鎖分子となる。本発明者らには、かかるアプローチによって、DNAが数メガベース(Mb)程度に増幅されることは当然予測され得る。本開示では、メガベースは、メガ塩基対も包含する。PolIIIホロ酵素の既知の合成速度に基づき、本発明者らには、複製フォークが、1Mb断片の場合でおよそ1Mb/1000秒、すなわち、およそ15〜20分間/サイクルの速度で進行すること予測され得る。
別の実施形態では、RPA増幅は、本明細書において「ネステッドRPA」という方法で行なわれ得る。希少な配列の検出における困難さは、標的配列に対して非標的が高比率であり得ることである。RPAが標的DNAと非標的DNAとの間を識別し、標的配列のみを増幅する能力は、感度の改善の重要な側面である。非標的と標的との識別は、プライマーおよび反応条件の特異性を反映する。反応が特異的であるほど、生成される特異的標的配列の相対量が多くなり、産物の検出が容易になる。したがって、特異性が増大すると、感度も増大し得る。
lsRPAおよびリーディング/ラギングRPAはともに、オリゴヌクレオチドプライマーを標的化するリコンビナーゼタンパク質の同様の使用に依存するが、これらは、増幅中に新たな娘二本鎖が形成される様式が異なる。リーディング/ラギングRPAでは、リーディング鎖およびラギング鎖が同時に合成される完全な複製フォークが確立され、その結果、2つの新たな二本鎖が同時に形成される。リーディング鎖RPA(lsRPA)では、リーディング鎖合成のみが起こり、その結果、合成により、産物として1つの二本鎖および1つの被置換単鎖DNAが生成される。
lsRPAにおける第2のDNA鎖の生成は、いくつかの方法の1つにおいて達成され得る。
lsRPA法は、いくつかの点でPCRと類似する。どちらの方法も、標的DNAにおいて互いに向かって指向される3’末端に関して配向される対のオリゴヌクレオチドプライマーを用い、どちらの方法も、反応生成物が後続のラウンドのDNA合成の標的として使用されることにより幾何級数的な増幅を達成する。しかしながら、反応組成において根本的な差がある。例えば、RPAでは、標的DNAは合成前で二本鎖であるが、PCRでは、鎖の熱分離後、単鎖である。RPAでは、DNA合成は、必然的に、鎖置換できるDNAポリメラーゼまたはポリメラーゼ複合体を用いなければならない。さらにまた、一部コピーされた鎖は、鋳型を介して被置換鎖と物理的に会合するため、ポリメラーゼが一時的に鋳型から解離した場合、新たな鎖の3’末端、および最終的に新たな鎖全体が、分枝点移動の作用またはバブル移動として知られる別の現象の作用によって失われるというリスクがある。これは、理想的に連続的合成能のポリメラーゼがRPA反応に使用されることを示唆する。また、集合性複製複合体が、ポリメラーゼの解離なしで容易に互いに通過することができない場合、一部のプロセッシング性のポリメラーゼは、一部の鋳型でのRPA反応を阻害し得ることを考慮することは重要である。まとめると、ポリメラーゼの理想的な選択は、具体的なRPA反応の正確な形式および目的、特に、増幅対象の産物のサイズに依存する。
第3の考慮事項は、如何にして、低温度で見られる異常プライマーアニーリングによって生じるノイズを抑えつつ、安定だが動的なリコンビナーゼ活性を確立するかである。これは、見かけ上、両立しないいくつかの要件が均衡する反応環境確立することを意味する。効率的なRPAのためには、リコンビナーゼタンパク質は、標的二本鎖DNAをスキャンするオリゴヌクレオチド/リコンビナーゼフィラメントに合成しながら、活性な状態を維持しなければならない。このような複合体はまた、鎖交換終了後、DNA合成を許容するために分解されなければならない。これは、単鎖DNAタンパク質高含有環境で起こるはずである。このようなタンパク質は、二次構造を融解することにより、異常オリゴヌクレオチド挙動を抑制しながら、組換えおよびDNA合成を刺激することが必要である。基本的に、リコンビナーゼおよび単鎖結合タンパク質は、オリゴヌクレオチド結合に対して競合状態にある。該単鎖結合タンパク質は、合成中の効率的な鎖置換を可能にするのに必要であるが、これらは、リコンビナーゼよりも単鎖DNAに対して高い親和性を有し、より協同的に結合するため、組換え活性が抑制される。
リーディング鎖RPA、リーディング鎖/ラギング鎖RPA、およびネステッドRPAの詳細は、上記した。このセクションでは、任意の上記の3つの方法のための試薬およびパラメータの選択を記載する。
核酸分子は、任意の生物に由来し得る。例えば、試料の微生物組成が、種々の微生物の核酸に指向される一連のRPA反応を用いることにより測定され得る。RPAは、微生物の検出に特に有用である。一実施形態では、病原体は、ウイルス、細菌、寄生虫および真菌から選択される。さらなる実施形態では、病原体は、インフルエンザ、風疹、水痘−帯状疱疹、肝炎A、肝炎B,他の肝炎ウイルス、単純疱疹、ポリオ、天然痘,ヒト免疫不全ウイルス、ワクシニア、狂犬病、エプスタイン−バー、レトロウイルス、およびライノウイルスから選択されるウイルスである。別の実施形態では、病原体は、大腸菌、ヒト結核菌、サルモネラ、クラミジアおよび連鎖球菌属から選択される細菌である。またさらなる実施形態では、病原体は、Plasmodium、Trypanosoma、Toxoplasma gondii、およびOnchocercaから選択される寄生虫である。しかしながら、本発明を上記の特定の属および/または種に限定することを意図しない。
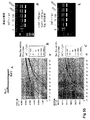
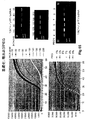
単鎖DNA結合タンパク質は、RPA反応に必要とされる。このようなタンパク質は単鎖DNAに結合し、二次構造を解き、放出鎖置換を助長し、分枝の移動を抑制する。RPAでは、それらの活性が、いくつかの異なる期(フェーズ)において必要とされる。本発明者らは、2種類の単鎖DNA結合タンパク質の大腸菌SSBおよびバクテリオファージT4 gp32の活性を調べた。T4 gp32は、本発明者らの手技で最も有用であることが示された。さらにまた、本発明者らは、ヘキサヒスチジン(ヒス)ペプチドタグをN末端またはC末端に含めることにより、このタンパク質のいくつかの異なる形態を作製し、先に記載されたいくつかの点変異を調べた。gp32改変体の活性を図21に概略的に示す。
T4 gp32タンパク質は、RPA反応に潜在的に有用ないくつかの特徴を有する。主なgp32は、比較的小さなDNA結合部位(8〜10ヌクレオチド)を有し、広範囲の塩濃度下で同様の結合特性を示し、モノマー間で高い(制限のない)協同性を示す(Scheerhagen et al.,J Biomol Struct Dyn.1986 Apr;3(5):887〜98;Kuil et al.,Biophys Chem.1988 Dec;32(2−3):211〜27)。対照的に、大腸菌SSBタンパク質は、塩濃度に伴って変化するいくつかの異なるDNA結合様式を有し、これらはすべて比較的大きなDNA結合部位(32、56または65ヌクレオチド)を有し(Ferrari et al.,J Mol Biol.1994 Feb 11;236(1):106〜23)、複雑な協同性挙動が存在する(LohmanおよびFerrari,Annu Rev Biochem.1994;63:527−70)。合成オリゴヌクレオチドを用いる場合、放出鎖の初期サイズは小さいため、本発明者らは、gp32タンパク質の特性はRPAに最適であろうと推論した。本発明者らは、N−末端ヒスタグを有するgp32(gp32(N))を発現させ、精製した。最初の実験では、本発明者らは、gp32(N)が、大腸菌recAリコンビナーゼと異種系で合わせた場合であっても、大腸菌SSBタンパク質と少なくとも同程度に機能することを見出した(図12)。これは、gp32がモノマーサブユニットの間で極めて高い協同性を示し、recAは、そのgp32の存在下でオリゴヌクレオチド結合に関して有効に競合し得ることは起こりえないようであったので、驚くべき結果である。しかしながら、本発明者らがgp32(N)の挙動を非タグ化gp32比較した場合、本発明者らは、これら2種類のタンパク質は、等価な挙動を示さないことを見出した。N−末端ヒスタグは、モノマー間の協同性に必要とされる場合、gp32タンパク質の「B」ドメインに直接隣接するため、本発明者らは、gp32(N)が協同性を減衰させたにちがいないと推論した。従って、本発明者らは、C−末端ヒスタグを有するgp32タンパク質(gp32(C))を作製し、そして3位のリシンからアラニンへの変化(K3→A)、または4位のアルギニンからグルタミンへの変化(R4→Q)もしくは4位のアルギニンからトレオニンへの変化いずれか(R4→T)を有する以前に公表された変異体に従うgp32(C)の点変異形態を、作製した(図21)。このような3種類の点変異タンパク質は、漸減する協同性を示す(図22)(Villemain,et al.J Biol Chem.2000 Oct 6;275(40):31496−504)。本発明者らは、これらのタンパク質の能力を2つの異なる濃度で、線状化した鋳型に対して、侵入/伸長反応を補助するために、バクテリオファージT4 uvsXタンパク質および大腸菌DNAポリメラーゼIのクレノウ断片との組合せで試験した(図22)。まず、本発明者らは、gp32(C)が、いずれの濃度でも他のgp32改変体よりもずっと少ない産物をもたらすこと(図22でgp32(N)と比較)およびこの産物が、gp32(N)とは対照的にもっぱら完全長であることに注目する。本発明者らは、これらの結果を一連の点変異対立遺伝子により得られたの結果と比較した場合、gp32(N)タンパク質が、gp32(C)R4→T(これは、協同性が有意に減衰されたと報告された)で得られたプロフィールに非常に類似することに注目した。これは、gp32(N)のN末端ヒスタグが、Bドメインの機能を点変異と同様の様式で妨げることを示唆する。
本発明者らは、前に、recAおよびgp32(N)を伴う非相同性系では、所与の鋳型からの1回より多いサイクルの侵入および伸長を可能にするためにポリエチレングリコールが必要とされることに注目した。ここでは、本発明者らは、この観察結果を合理的に説明し、なぜ、複数回のDNA末端標的化が、特異的型の単鎖DNA結合タンパク質を必要とするのかを説明するためのモデルについて記載する。gp32活性が、鎖交換プロセス中に放出鎖を安定化させるために必要であることは明白である。これが起こらなければ、リコンビナーゼが分解されるにつれて、放出鎖がその相補鎖と再ハイブリダイズし、侵入オリゴヌクレオチドと置換される。鎖交換後、単鎖DNA結合タンパク質に対して異なる要件を出し得る2つの状態のうちの1つ状態で、放出鎖が存在し得る。これらの2つの状態は、一本鎖検索DNAと二本鎖標的DNAとの間の関係の結果として生じる。組換え事象が線状二本鎖DNAの一端まで延び得、そして放出鎖がその相補鎖から一端で完全に除去されることが可能ならば、放出鎖は、一端において非拘束的となる。この非拘束的状態では、インカミングDNA鎖およびその相補鎖を含む新規二本鎖が、巻き戻されてB形DNAを形成する。このことは、対合形成反応の間に標的DNAが侵入リコンビナーゼフィラメントの活性によって巻かれた状況にあるため、必要である。非拘束的放出鎖は、単鎖DNA結合タンパク質に容易に結合し得、これにより、分枝の移動が発生し、侵入鎖が除去されることが抑制される。
単鎖DNA結合タンパク質は、リコンビナーゼ負荷が一貫して起こり得るように二次構造を解くことにより、リコンビナーゼ負荷のための単鎖DNAの調製を補助し得る。従って、単鎖DNA結合タンパク質の解く活性は望ましく、また、プライマーの非特異的アニーリングを止めること(silencing)において1つの役割を果たす。しかしながら、このことにもかかわらず、過剰のレベルのタンパク質および過剰の協同性は、侵入に利用可能なリコンビナーゼ負荷フィラメントの数を有意に減少させ得る。
単鎖DNA結合タンパク質は、リコンビナーゼがバラバラになると、放出鎖を収集し、インカミングオリゴヌクレオチドの自発的排出を抑制する。
並行接合の不安定性とは、組み込まれた配列において侵入が起こることを意味し、オリゴヌクレオチド末端が二本鎖末端と端を揃えた(flush)(増幅のほとんどのサイクル中で起こり得る)場合を含む。これは、多くの状況で有意な協同活性が必要とされ得ることを意味する。一般に、このフェーズのこの反応は、高度に協同性の単鎖DNA結合タンパク質の余剰分から恩恵を受ける。
単鎖DNA結合タンパク質は、DNA合成中に形成される、置き換えられる鎖に結合する。フェーズ2のように、この置き換えられる鎖は、非拘束であっても位相的に拘束されていてもよく、このような2つの環境は、単鎖DNA結合タンパク質に対して異なる要求を課す。
RPA反応の特定の構成では、置換された単鎖の放出鎖は、パートナーオリゴヌクレオチドにハイブリダイズして後続の新たな二重鎖の生成を可能にしなければならない。多くの単鎖DNA結合タンパク質は、相補鎖がアニーリングするの抑制するが、T4 gp32タンパク質は、相補的DNAの再アニーリングを助長する。従って、バクテリオファージT4 gp32タンパク質は、このフェーズに理想的なタンパク質である。
RPAで使用される比較的短い合成オリゴヌクレオチドプライマーを用い、どれだけ有効にrecAまたは他のリコンビナーゼが使用され得るかを支持する証拠は、ほとんど発表されていない。また、かかる短いDNAオリゴヌクレオチドがリコンビナーゼにより能動的に負荷された状態で維持される、安定な反応環境が形成され得るか否かは明らかでない。recAを用いて行なわれる研究のほとんどでは、比較的大きな基質、例えば、単鎖形態および二本鎖形態のバクテリオファージM13ゲノム(数千残基)を、ドナーDNAおよびアクセプターDNAとして用いる。リコンビナーゼ活性の実験アッセイは、多くの場合、中間体の形成、または完結した組換え事象から構成され、電気泳動移動または電子顕微鏡によって測定される(Harris LD,Griffith J.J Biol Chem.1987 Jul 5;262(19):9285−92)。いくつかの実験では、短いオリゴヌクレオチドの使用が記載されている。15ヌクレオチドほどの短い配列は、非加水分解性補因子アナログATP−γ−Sの存在下で、recAとともに機能的相同性検索複合体を合成することが示されているが、短いオリゴヌクレオチドとATPとを組み合わせた調査は、明瞭でない(Hsieh P,Camerini−Otero CS,Camerini−Otero RD.Proc Natl Acad Sci U S A.1992 Jul 15;89(14):6492−6)。リコンビナーゼの相同性検索機能は、鎖交換を終了し侵入複合体から解放して、ポリメラーゼなどの他のDNA代謝タンパク質による接近を可能にするのには、必ずしも充分でとは限らない。実際、研究により、recAは、検索に必要とされる15ヌクレオチドよりかなり長いオリゴヌクレオチド長で、低ATP加水分解速度(DNA結合活性と機能的リコンビナーゼ活性との組合せの有用なインジケータ)と高加水分解速度との間での移行を示すことが示されている。さらにまた、ヌクレオチド補因子の型は、かかる加水分解の移行が起こる長さに影響するようである(Katz FS,Bryant FR.Biochemistry.2001 Sep 18;40(37):11082−9)。また、バクテリオファージT4 リコンビナーゼuvsXは、短いオリゴヌクレオチドに対して種々の特性を示すことが示されており、塩基組成に対していくらか感受性を示す(Formosa T,Alberts BM.J Biol Chem.1986 May 5;261(13):6107−18)。このことにも関わらず、uvsXおよびrecAはともに、ATPなどの加水分解性ヌクレオチドの存在下で、ほぼ30塩基対以上の単鎖基質により組換え事象行なうことができ、これは、かかる短い合成標的化オリゴヌクレオチドの使用が合理的であることを示す(Salinas F,Jiang H,Kodadek T.J Biol Chem.1995 Mar 10;270(10):5181−6;Formosa T,Alberts BM.J Biol Chem.1986 May 5;261(13):6107−18)。
(1)組成および配列)
インビトロDNA合成反応での使用、特にPCRにおける使用のためのオリゴヌクレオチドを設計するためのソフトウェアは、充分確立されている。RPA法の考慮事項は類似であり、オリゴヌクレオチドの融解温度の最適化、オリゴヌクレオチド内のヘアピン形成の回避、および所与の反応に存在する他のオリゴヌクレオチドとの相補性に対する選択が挙げられる。RPAは、比較的低温度で行なわれるため、かかる考慮事項は潜在的により重要になる。本発明者らは、RPA反応において伸長されたプライマー産物の蓄積を観察し、これは明らかに、鋳型非依存的でありそして使用するプライマーの組合せに依存的である。生成される異常産物のサイズは、これらがプライマー二量体であるか、または単一のオリゴヌクレオチドの自己プライミングの結果を示す。このような好ましくないプライマーアーティファクトは、PCRなどの他の方法でよく知られている。従って、好ましくない副反応が回避されるオリゴヌクレオチドプライマー対を設計することは重要である。本発明者らは、ヘアピンを形成し得るオリゴヌクレオチドは、RPA反応において誤って自己プライミングし得ることを観察した。
また、オリゴヌクレオチドの3’末端で異常伸長を阻止することも、ハイブリッドの形成に関して標的化オリゴヌクレオチドの3’領域と効率的に競合し得る短いコンペティターオリゴヌクレオチドを設計し、これを含めることによって達成され得る。このようなアプローチは、種々の様式で構成され得る。
RPAに使用され得るDNAポリメラーゼはたくさんある。しかしながら、所与の適用に最適なRPA形式を設計する際に考慮すべきいくつかの基準がある。本発明者らは、RPA反応において活性を有するいくつかの異なるポリメラーゼを同定し、どの特性が種々の環境に特異的利点をもたらすのかを推測した。刺激的な結論の1つは、非相同性系由来のポリメラーゼが有効に使用され得ることである。本発明者らは、以下に、RPAに最も関連するポリメラーゼ活性を記載する。
ポリメラーゼ連続的合成能(プロセッシビティー)は、DNA鋳型との各個々の相互作用において触媒された、組込み事象の代表的な数値として測定される。分子生物学適用において使用されているポリメラーゼ酵素の多くは、多くの場合、その主な役割がDNA修復およびオカザキ断片の連続合成である大腸菌DNAポリメラーゼIの機能的アナログであるため、高度に連続合成的ではない。連続的合成能は、RPAにとって、PCRよりも不可欠な考慮事項である。RPAでは二本鎖鋳型を使用するため、分配性ポリメラーゼは、接合を有する部分的にコピーされた鎖をもたらし、これは、分枝の移動によって排出され得る。また、本発明者らは、バブル移動は、さまざまなRPA構成で起こり得ることを示す証拠を有する。バブル移動において、鋳型DNAの親鎖は、複製複合体のすぐ後ろで再ハイブリダイズし、新たに合成された鎖の放出をもたらし、この鎖は、元の組換え事象の放出鎖の代わりに単鎖DNAとして遊離している。この再ハイブリダイゼーションは、以前に、T4 組換え/DNA合成系において報告されており、uvsXリコンビナーゼによる放出鎖の被覆および後続の再侵入を伴うと考えられている。あるいはまた、非協同性単鎖DNA結合タンパク質の存在下で、モノマーは放出鎖の一方の末端から徐々に失われ得、5’−3’方向に進む進行的な分枝の移動をもたらし得、複製複合体に追従し得る。
多くのDNAポリメラーゼは3’−5’エキソヌクレアーゼ活性を有し、一部はまた、5’−3’エキソヌクレアーゼ活性も有するが、これは、ポリメラーゼが前方に移動するにつれて、置換ではなく、一方のDNA鎖の消化が鎖進行的にもたらされるため、おそらくRPAにおいて好ましくない。3’−5’エキソヌクレアーゼは、潜在的に利点を有すると同時に明白な不都合点を有する。一方では、3’−5’エキソヌクレアーゼ活性は、複製反応の忠実度を増大させ、また、組込み間違いの地点でのポリメラーゼの失速を抑制し得る。高忠実度の増幅は多くのDNA適用に望ましい。3’−5’エキソヌクレアーゼ活性はまた、大きなDNA断片の増幅に適切であり得る。組込み間違いによる失速は、効率的な増幅を阻害し得る。
侵入後に形成される組換え中間体は、DNAポリメラーゼに接近可能でなければならない。標的化オリゴヌクレオチドの3’末端付近の構造は、それ以外は単鎖DNAとハイブリダイズした(PCRの状況である)オリゴヌクレオチドの3’末端と等価ではない。その代わり、侵入オリゴヌクレオチドの3’末端のすぐ下流またはやや下流で鋳型鎖にハイブリダイズする放出鎖が、ポリメラーゼ負荷をブロックし得る。さらに、放出鎖が拘束されているか拘束されていないかは、ある種のポリメラーゼが成功裏に負荷を与える能力に影響を与え得る。特定のポリメラーゼがこのような状況において有効に機能を果たし得るか否かは、実験的により取り組まなければならない。本発明者らは、大腸菌DNAポリメラーゼIのクレノウ断片、およびBacillus stearothermophilusから精製されたBst DNAポリメラーゼは、かかる組換え中間体に負荷を与え、伸長させ得ることを見出している。また、T4 ddaヘリカーゼおよびT4 gp41ヘリカーゼなどのヘリカーゼが、組換え中間体を連続合成し、鋳型と放出鎖を交換事象の下流で分離させ、他のポリメラーゼが使用されるのを可能にする機能を果たし得るが、これらのヘリカーゼもまた、RPAの他の側面を妨げ得る(下記参照)。最後に、RPA反応において相乗的に作用するポリメラーゼの混合物(例えば、一方のポリメラーゼが組換え中間体の3’末端への接近に効率的であり、他方は、連続合成性の鎖置換合成活性を有する)用いることは有益であり得る。
本発明者らは、異種系由来の酵素性成分を種々のRPA形式において一緒に有効に組合せ得ることを示した。例えば、大腸菌DNAポリメラーゼIのクレノウ断片、Bacillus stearothermophilusのBst DNAポリメラーゼ、およびBacillus subtilisポリメラーゼIのラージフラグメントはいずれも、バクテリオファージT4 gp32タンパク質の存在下で、バクテリオファージT4のuvsXリコンビナーゼにより生成される組換え産物を伸長させ得る。これは、大腸菌Pol Iと類似するポリメラーゼの類型が、一般的にRPA反応において有効であることを示唆し、これは、それらの修復活性に必要とされる組換え中間体への接近と組合せた、それらの鎖置換酵素としての異なる特性を反映し得る。さらにまた、組換え中間体の伸長が、広く異なる構造群に由来するポリメラーゼによって容易に媒介され得ることは不確実である。バックファイヤー合成によって安定化される末端指向組換え中間体により初期に示されたにもかかわらず、本発明者らは、φ−29 ポリメラーゼを用いてRPA反応を誘発することはできなかった。これは、この酵素が組換え中間体上に容易に負荷され得ないことを反映し得る。同じ生物由来のタンパク質を一緒に用いた場合、さらなる相乗効果があり得る。例えば、バクテリオファージT4成分(例えば、uvsYおよびuvsX、ならびにgp32)の間に物理的相互作用があることが知られている。このことは、他の成分に拡張され得、例えば、T4ポリメラーゼは、gp41ヘリカーゼと、物理的にgp32と機能的に相互作用する(FormosaおよびAlberts PNAS 80,2442−2446,1983)。しかしながら、RPA効率を向上させるために同じ生物由来の成分を使用することは、一見魅力的であるにもかかわらず、本発明者らは、T4ポリメラーゼによる連続合成性のDNA合成に用いられる一部のヘリカーゼは、本発明で確立した有効なRPA環境と容易には組合せられ得ないことを見出した。初期の実験により、T4 ddaヘリカーゼを含めることは、RPA環境においてオリゴヌクレオチドの被覆を崩壊させ、過剰のプライマーノイズをもたらし、RPA反応の感度を妨げることが示された。これは、以前に公表された報告[差込参照:JBC.1991 May 25;266(15):9712−8 Inhibition of protein−mediated homologous paring byb a DNA helicase.Kodadek T]と一致する。
いくつかの形式のRPAでは、例えば、大きなDNA産物が所望される場合、連続合成性のポリメラーゼが最適な選択であり得る。しかしながら、この条件では、複製複合体が同じ鋳型で互いに集中する有意な可能性がある。複製複合体は、直接対面して(head−to head)動かなくなる状態になり、その結果、いずれも通過することができないという危険がある。この状況で最も有用なのは、連続合成性で、かつ衝突を解決できるポリメラーゼであり、φ29 DNAポリメラーゼについて実証されているとおりである(Elias−Arnanz M,Salas M.EMBO J.1997 Sep 15;16(18):5775−83)。あるいはまた、かなり高頻度に解離させる分配性能力と組合せた有用な複製実行を可能にするための、連続的合成能の理想的なレベルの微細だが重要な均衡も、この問題を解決し得る。
本発明者らは、大腸菌recAタンパク質およびバクテリオファージT4 uvsXタンパク質の両方をRPA実験においてアッセイした。これらのタンパク質はともに、一部の限定的なタンパク質配列相同性を共有し、共通の先祖から進化したと考えられる。これらのタンパク質の核タンパク質フィラメントの結晶学および電子顕微鏡研究では、ATPおよびADPが結合した両方の状態で形成されたらせんのピッチの点で保存されたフィラメント構造が示され、これは、それらの作用機構において顕著な類似性を示す。さらにまた、すべての原核生物は、recAに高度に相同なタンパク質を有し、このことは、リコンビナーゼの主要な活性が、進化を通じて保存されていることを示す。従って、1つのリコンビナーゼからわかることは、別のものに当てはまり得、または置き換えられ得る。
RPAを強化するため、反応を、充分な数の活性な被覆された相同性検索リコンビナーゼ/DNAフィラメントを供給するように構成することは重要である。また、相同性検索の終了後、フィラメントは、効率的に分解されるか、あるいはそれ以外に処理され、DNAポリメラーゼおよび他の成分の負荷を可能にしなければばらない。また、オリゴヌクレオチド融解の助長、および置換された放出鎖の収集の両方のために、充分な量の単鎖DNA結合タンパク質が存在することは不可欠である。最終的に、強力なRPAでは、連続合成性の鎖置換DNA合成が必要となる。このような要件の基礎をなすことは、リコンビナーゼと単鎖DNA結合タンパク質の二者間の競合である。
いくつかの特定の反応成分は、RPA反応有効性に対して有意な影響を有する。
ポリエチレングリコール(PEG)は、組換え/DNA合成に対して著しい効果を有する。第1に、本発明者らは、PEGが、例えば、recAをgp32(N)と組合せた場合に起こる複数回の侵入/伸長サイクルの回数に影響することを見出した。また、本発明者らは、PEGが、いくつかの異なる様式で構成した増幅反応を刺激することを見出した(図15、実施例3)。また、いくつかの構成において、PEGにより、形成される産物長さ分布が改変されることがわかった(図28)。まとめると、ポリエチレングリコール、およびおそらく他の同様のクラウディング剤は、gp32とリコンビナーゼの協同性に影響し、ポリメラーゼの連続的合成能に影響し、溶液中でのハイブリダイゼーション速度およびオリゴヌクレオチドの挙動に影響し得る。これらは、反応体を相分配することおよび/またはフラクタル様反応速度論の進行を引き起こすことにより、細胞様環境に入れ得る。さらにまた、ポリエチレングリコールの鎖長は重要であるようである。試験したもののうち、平均分子量1450および15,000〜20,000のPEG(PEG「化合物」)が最良の結果をもたらすことがわかった。gp32機能、特に、協同性が減衰されたgp32改変体の機能におけるPEGによる助長は、上記に詳述した。また、PEGはリコンビナーゼ負荷フィラメントの安定性を増大させ得、増大された持続性により、RPA有効性が増大し得る。最も重要なことには、本発明者らは、ポリエチレングリコール(PEG 1450では不十分で、PEG化合物では非常に効率的)を用いずには、微量の試料を検出可能なレベルまで増幅できる増幅条件の確立を完全にはできていない。おそらく、他の因子もRPA反応を刺激し得る。しかしながら、この効果は、ポリビニルアルコールを用いた予備実験では明白でなかった。従って、ポリエチレングリコール、より詳しくはPEGの特定の改変体の特異的特徴は必須であり得る。
ATP再生系は、リコンビナーゼが、核酸に結合したとき、極めて高いATP加水分解速度を有するので、持続性の組換え反応を可能にするのに非常に重要である。特に、uvsXタンパク質はrecAより10〜20倍高い加水分解速度を有し、1分当たりモノマー当たりATP 200分子を消費し得る。いくつかの系が利用可能であり、本発明者らは、クレアチンキナーゼ/クレアチンリン酸系を常套的に使用した。uvsXを用いる場合、生成されるAMPはATPに変換されなければならない。本発明者らは、AMP1分子とATP1分子とを2つのADP分子に変換するニワトリのミオキナーゼを使用した。次いで、ADPをATPに、クレアチンキナーゼ/クレアチンリン酸系を用いて変換する。ATPの不十分な再生は反応速度を低下させ、検出可能なレベルの産物が蓄積される前に反応が停止することがあり得る。
DNA合成中、およびuvsXを用いた場合にATPがAMP+PPiに加水分解されるため、ピロリン酸塩(PPi)が蓄積される。反応物中のPPi蓄積は、いくつかの有害な結果を有し得る。有意なピロリン酸塩蓄積は、許容され得ない速度の加ピロリン酸分解をもたらし、それにより、ポリメラーゼの合成反応が逆方向に誘発され、最も3’側のヌクレオチドを二本鎖鋳型から除去する。これは、好適な二本鎖領域が現れて迅速な伸長が可能になるまで、ポリメラーゼの編集活性が3’−末端を元にトリミングする傾向にあるため、許容され得ないレベルのプライマーノイズ、または放出鎖の好ましくない自己プライミングのレベルの上昇をもたらし得る。さらに、ピロホスファターゼ蓄積は、リコンビナーゼの阻害およびポリメラーゼ合成反応の遅延をもたらす。
RPAは、高い感度および特異性を示す。しかしながら、標的をまったく含有しない試料は、多くの場合、プライマーにのみ由来する産物を生成させる。かかる現象は、他の方法では珍しいことではない。例えば、PCR法では、最終的に非特異的産物、例えば、いわゆる「プライマー二量体」が生成される。RPAは、小さなアンプリコンがインキュベーション期間内で多数回の倍増を達成するのを可能にするサイクル制御がないため、いくぶん、かかるプライマー関連性アーティファクトがもたらされる傾向がある。反応産物の電気泳動により、シグナルとノイズの容易な分離が可能になる。しかしながら、簡単な非実験室用の診断用産物では、他のアプローチを採用しなければならない。従って、本発明者らは、かかる非ゲル診断試験を、充分な信号対ノイズで機能させるため、および検出を容易に行なうためのアプローチを開示する。本発明者らはまた、周囲温度で多くの日数保存し得る(非実験室での容易な使用のための必要条件)反応凍結乾燥物の組成物を開示する。
DNA増幅系は、一般的に、真の標的から開始されないDNAの増幅が起こり得るという事実に対処する。この「ノイズ」は、特に、見かけ上標的が少ない条件下では、許容される感度および増幅の評価方法に対して制限を与え得る。初期のRPA構成は、優れた感度を提供するが、本発明者らは、リコンビナーゼを主とする系の新規な特性により、特異性を改善するためのこれまで利用されていないアプローチ、および新規な産物検出スキームの開発に適合性となると考える。
PCRまたは従来のハイブリダーゼーションを伴う他の方法とは異なり、RPAが、活性な相同性検索複合体としてATP加水分解を行なう伸長された核タンパク質フィラメントの形成に依存性であることは、オリゴヌクレオチド活性を決定するパラメータに対して、以前、インビトロ増幅反応では見られなかった様式で影響を及ぼし得る。本発明者らは、オリゴヌクレオチド活性の上記の30残基未満での急速な低下が、組換え速度の低下を反映していること、そしてこれのことがおそらくは、別途、大腸菌recAリコンビナーゼの場合のATP加水分解の長さ依存性に関して行なった所見(Bianco PRおよびWeinstock GM)に対応することを、示す。特に、短い配列の長さおよび組成は、ATP加水分解に影響することが示された。ほぼ30残基以上は、最大ATP加水分解(1モルのDNA結合recAに対するATPのモル数)に必要とされ、この場合、recAは、合成オリゴヌクレオチドに結合されており、より短い配列は、約15残基まで、存在しなくなるまで加水分解速度の著しい低下を示す。本発明者らは、低増幅挙動の観察結果を、ATP加水分解に関する以前のデータと関連させた。なぜなら、核タンパク質組換え活性がATP加水分解速度によって影響されると考える明白な論理的根拠があるためである。ある意味では、本発明者らが観察した最低速度のオリゴヌクレオチドは、非加水分解性ATP−γ−Sと結合されたものであって、交換終了(分解されたらせん接合の形成)の速度がほぼゼロのものである(Riddles PWおよびLehman IR)。本発明者らはまた、ATP−γ−SがRPA反応を有効に害すること(Piepenburg et al.)を証明した。本発明者らは、核タンパク質フィラメントにおける高速のATP加水分解は「高速」プライマーの活性を支持し、低速のATP加水分解は「低速」プライマーの活性を支持する。これらは、主に、単位時間内に完了する完全な組換え事象の平均数を異にする。単純に解釈すると、ATP−加水分解は、フィラメントの動的活性に必要とされる。30残基未満のオリゴヌクレオチドは、より安定な、非動的となり、最終的に、ATP−γ−Sを用いた場合に生じるロックされた状態になり、交換を完了できなくなる(「低速」オリゴヌクレオチドは、配列を容易に位置づけ得るが、容易に分解されて完了およびポリメラーゼ接近を可能にすることができない)。
なぜ低活性な「低速」オリゴヌクレオチドを選択することに何らかの利点があり得るのかを理解するため、本発明者らは、どのようにしてプライマー「体操」ノイズが生じ得るのかを探究することが必要である。短縮したオリゴヌクレオチドはノイズを、特異的シグナルよりも高速で低減し得るのか?真の標的およびノイズ産物の両方で見られる増幅のうちの幾何級数的フェーズとは異なり、出発プライマー由来のノイズ性標的の初期形成フェーズは、おそらくより複雑であり、おそらくかなり低速である。図43に、本発明者らは、個々のプライマーが幾何級数的な増幅能力を有する構造に変換された状態となり得る2つの一般的な機構を詳細に示した。図示したどちらのストラテジーも、単一のプライマー種由来のアンプリコンの形成を示す。どちらの場合も、第1の工程は、3’−末端での短いヘアピンの形成によるオリゴヌクレオチドの自己プライミングを伴なう。ストラテジー1では、生じるヘアピンは、有意なミスマッチのため、幾何級数的増加(geometric)フェーズにおいて容易に増幅しない。幾何級数的増加フェーズのアンプリコンになる代わりに、これらは、ストラテジー2に記載するものとより類似した工程により進行する必要があり得る。この場合、第1の自己プライミング事象の後、元のオリゴヌクレオチドの5’末端に対する逆相補体が関与する第2のものが続く。かかるモデル作製からいくつかの簡単な見解がある。第1は、2つ型の事象が起こらなければならないことであり、オリゴヌクレオチドの3’末端からの伸長がなければならず、次いで、第2の同様の3’ヘアピンおよび伸長がなければならず、これは、元のオリゴの5’末端に対する逆相補体が関与する。幾何級数的フェーズを進行する第1の事象の非常に希少で不安定な性質は、大部分のオリゴが、SSBまたはリコンビナーゼ(これらは、ssDNAに結合したときDNA融解活性を有する)のいずれかで被覆された場合、最も強く阻害されることになることに注意されたい。逆に、自発的に分解する動的なリコンビナーゼフィラメントは、オリゴヌクレオチドの全部または一部が被覆されていない時間枠を作りだす。従って、この非常に稀な限定的工程は、プライマーATP加水分解速度に対して、動態性に対して、および外挿によるオリゴヌクレオチド長に対して、感度がよいものであり得る。従って、オリゴヌクレオチドを短縮することは、特に有効でノイズを抑制し得、真の標的増幅速度の低速化を正当化してその増幅速度の低速化を達成するためのするのに充分である。
上記の4つの観察は、ある程度、直接的なオリゴ設計原理に有用である。
最適なプライマー長は、およそ30残基であり得、場合によっては、数残基短いものが優れた信号対ノイズ比をもたらす。平均プライマー長を短くするとノイズ生成が減少し、26〜28量体を用いると、完全に非存在となるようである(図45)。標的の増幅もまた減少するが、本発明者らは、ノイズは、シグナルよりも急速に減少するのではないかと考える。どのようにしてノイズが生じ得るかの以前の解析では、本発明者らは、なぜ、プライマーノイズが正しいアンプリコンよりも、組換え速度の減少によって影響され得るかには、いくつかの理由があると結論づけている。
最も5’側残基の伸長部以外は、同一の配列を有するオリゴヌクレオチドの活性を比較することにより、本発明者らは、オリゴヌクレオチドノイズの程度の有意な多様性が観察されることに注目した(図42、45、47を参照)。可能性の1つは、これが、純粋に、上記のように、核タンパク質活性に対する長さの多様性の効果を反映していることである。しかしながら、これは、比較対象のプライマーがすべて30残基以上である場合での唯一の理由であり得る。例えば、図42では、オリゴヌクレオチドNEST−1およびJlは、Jlがいくつかのさらなる5’残基を含有する以外は同一である。よりノイズ性,であるどころか、このオリゴヌクレオチドはノイズ性が低いが、非常に良好に増幅する。この場合などに基づき、本発明者らは、2つの他の同様の機構を提案する。第1には、オリゴヌクレオチドを指数関数的に増幅可能にすることは、最も5’側配列の逆相補的な鎖によるプライミング事象を必要とし得、この事象は、不可欠に、5’末端の塩基の正確な配列に依存する。これは、最も5’側塩基の変化がノイズ生成の有意な多様性をもたらし得ることを意味する。第2に、オリゴヌクレオチドが30塩基より大きい場合であっても、5’配列の塩基組成でさえ、プライマーの組換え活性に影響を及ぼし得る。これを補助する証拠は、伸長鎖ホモポリマーからなる5’タグをプライマーに添加すると、その結果が観察される実験に由来する。図47は、1回のランのC残基またはG残基がオリゴヌクレオチドJlおよびK2に付加され、続いて、すべての生じ得る修飾オリゴヌクレオチドおよび非修飾オリゴヌクレオチドを、単独または生じ得る組合せで含有する増幅反応が確立された実験を示す。驚くべきことに、既に、非常に感受性で特異的であると決定されているこのようなオリゴヌクレオチドは、このような特定の伸長鎖ホモポリマーを添加することによって改善されなかった。代わりに、1回のランのシトシンを5’末端に添加すると、これは、単独でインキュベートした場合、非特異的産物の産生の増加をもたらすが、グアノシンの伸長鎖は逆の効果を有し、単独で放置すると、オリゴヌクレオチドが非常に静的となることが観察された。また、元の親の非修飾オリゴヌクレオチドの組合せとは別に、見かけ上、成功裏に正しい産物を生成する唯一の対合は、2つのシトシンテイルオリゴヌクレオチド(すなわち、最もノイズ性のオリゴヌクレオチド)の組合せであった。このような観察を正当化し得る説明の1つは、5’末端のシトシンの伸長鎖が、結果として生じる核タンパク質フィラメントをより活性にするが、グアノシンはあまりそうしないことである。まとめると、長さなどのオリゴヌクレオチド組成は、速度挙動に影響を及ぼし得、さらなる無関係の配列5’末端は、この点において有意な影響を有し得る。最適なプライマーは、5’配列設計に対する合理的なアプローチにより見い出され得る。いくつかの異なる最も5’側塩基も試行され得、最良の選択が使用され得る。さらに、タグがその配列が標的に無関連である5’末端に配置され得ることがあり得るが、逆相補鎖からのヘアピンプライミングのスナップバックに対する抵抗性を付与する。多くのオリゴヌクレオチドに作用する最適な配列の決定が可能であり得る。反応成分の存在下でオリゴヌクレオチドのATP加水分解速度の測定もまた、最適プライマー対の設計において有用であること証明し得、すべてが同一の増幅速度挙動を共有するプライマーミックスの作製可能にし得る。長さなどの、このような観察結果および改善は、これらが、リコンビナーゼ誘発性増幅系の特異的特徴および根本的な生化学に依存するため、他の系では明白ではないだろう。
ロックされた核酸(LNA)は、連鎖がリボースの糖環の2’および4’炭素が遺伝子操作されたヌクレオチドである。かかる糖を、最も特別にはまさに3’位置に含有するオリゴヌクレオチドは、PCRアッセイ成功裏に使用されており、正しい3’塩基対合の識別を有意に改善することが示されている(Vester B. and Wengel J.を参照)。本発明者らは、LNA残基は、オリゴヌクレオチド内の多すぎる位置に存在しないならば、RPA系により耐容性であり得ると予測した。これらは、PCR増幅の特異性を増加させることが知られているため、RPA系におけるシグナル−ノイズを良好に改善し得る。
プライマーからの組換えおよび/または伸長の速度を、それらの長さ、塩基組成および分布および修飾糖(あるいは他の主鎖修飾)の使用に基づいて制御するできることは、最適信号対ノイズ特性により増幅反応により設計する機構を示す。本発明者らが想定している好都合に使用されるオリゴヌクレオチド組合せストラテジーには、2つの一般的な方法がある。第1は、単一のチューブネスティングが起こるストラテジーであり、第2のものは、より従来型のオリゴヌクレオチドと、真のアンプリコンが容易に形成されるが、ノイズ性アンプリコンは単一のオリゴヌクレオチドしか伴なわないように対合形成された非ノイズ性オリゴヌクレオチドを用いるものである。
ネスティングは、第1の増幅をアウタープライマー対を用いて行ない、次いで、第2の増幅をインナープライマー対を用いて行なうことにより、改善された感度および信号対ノイズが可能となるプロセスである。その最もよく見られる状況では、このアプローチは、PCR法との組合せで使用されている。主に、第1のラウンドでは、標的がランダムDNAより効率的に増幅され、その結果、第1の増幅後、バックグラウンドに対して明白なアンプリコンが得られるには均一性が不充分であっても、なお、比較的非常に高含有状態であるため、第1のものに対して内部であるプライマーによる第2の増幅では、非常に明瞭な結果が容易にもたらされる。実際には、これは、通常、1つの増幅の実施、次いで、小部分を新たなチューブへの取り出し、および内部「ネステッド」プライマーによる第2の増幅の実施に依存する。
本発明者らが得た実験データは、プライマー誘発性スミアまたはラダーの大部分は、一方のプライマー種のみに由来する配列を含有することを示す(図46を参照)。真実ならば、この事実単独で、反応に使用した2種類のプライマーが産物に物理的に会合しているか否かを単に調べることにより、シグナルをノイズと識別する簡単なアプローチが示される。従って、本発明者らは、これが、非常に静的なオリゴヌクレオチドを高速の(よりノイズ性)オリゴヌクレオチドと組み合わせることによりさらに改善され得ることを示す。静的なオリゴヌクレオチド、例えば、短いオリゴヌクレオチドは、よりノイズ性のものとともに存在している場合、依然として効率的に機能し得る(ハイブリダーゼーション反応において機能し得るものとする)(図52を参照)。しかしながら、これらが、単に、その反応速度論がリコンビナーゼ促進型ノイズ増幅と非常に異なる正常なハイブリダーゼーションにより増幅に関与しているだけの場合、ノイズには関与しない状態で維持され得、より高速なオリゴヌクレオチド単独が関与する。
プライマーが上記の体操単独に主として関わる可能性は、増幅が効率的に起こったか否かを調べるためのを簡単なアプローチを示す。簡単な非ゲル検出形式の1つは、一方が色素もしくは酵素または他の容易に検出可能な基で標識されたオリゴヌクレオチド、および他方が固定化可能な基、例えば、ビオチンで標識されたものを用いるものであり得る。(図5Oパート1を参照)。従って、主反応の反応期の前、その間、またはその後に、固定化可能なオリゴヌクレオチドを表面に固定化させ得、反応の最後に、この表面を適切なバッファーで洗浄し得る。他方の標識されたプライマーが固定化可能なプライマーと同時会合した場合(これは、容易に測定され得る)、産物の増幅が起こっている。
非実験室用または患者の近くでの診断用および法医学的試験を構成するため、周囲安定性試薬を提供することが必要である。明白な方法の1つは、これを、反応物を凍結乾燥することにより達成することであり、試料DNA、あるいは、独立して安定であり、試料とともに添加され得る任意の他の成分(例えば、試料核酸を溶解するために使用されるバッファー)のみが除外される。
DNA増幅反応の反応速度論解析を行なうことが可能なことは、同様の反応の単に末端−点解析と比べ、非常に大きな実用性を提供する。1つには、これは、試料中に存在するDNAまたはRNAのコピー数の測定を可能にし、研究、医療、および環境試験での適用に多くの用途を有する。直接的な定量適用に加え、動的反応産物検出は、他の問題に対する優れた解決策、例えば、改変され蓄積反応速度論によって一ヌクレオチド多型性対立遺伝子の識別を可能になることなど、および非ゲル系形式標的の有無を評価する機構をもたらす。
RPAは、多目的な方法であるが、反応においてリコンビナーゼが活性な場合に、正確に制御する特徴を組込むことによって改善され得る。かかる制御は、ケージ化ATPの光分解によるATPの周期的な解放によって得られ得る。あるいはまた、ATPまたは他のヌクレオシド三リン酸の反応濃度を、反復的な手動添加または生化学的オシレータの使用によって周期的に調節し得る。
一方向RPA反応は、単一のプライマー標的部位から誘発され得る。対向プライマーの非存在下では、かかる反応では単鎖DNAが生成される。本発明者らは、3’ロックされた核酸(LNA) [Di Giusto DA,King GC(2004) Strong posisional preference in the interaction of LNA oligonucleotides with DNA polymerase and proofreading exonuclease activities:implications for genotyping assays.Nucl Acids Res 32(3):e32]ヌクレオチドを有するオリゴヌクレオチドは、特定のポリメラーゼ、例えば、Bacillus stearothermophilusおよびBacillus subtilisポリメラーゼIによるリコンビナーゼ媒介性伸長のプライマーとしての機能を果たし得ないが、他のポリメラーゼ、例えば、E.coliクレノウ断片プライマーとしての機能を果たすことを見出し、これは、かかる3’LNAキャッププライマーが、標的単鎖DNAにハイブリダイズすると、ポリメラーゼ伸長反応においてプライマーとしての機能を果たし得ることを示す以前のデータと一致する。本発明者らはまた、すべてのポリメラーゼが組換え中間体から開始できるわけではないようであり、おそらく、一部のポリメラーゼでは、単鎖DNAのより長い伸長鎖が、プライマーと結合していることが必要とされることを反映していることを見出した。例えば、本発明者らは、φ−29ポリメラーゼは、組換え中間体からの合成を開始できないことを見出した。しかしながら、また、本発明者らは、これが、3’LNAキャッププライマーから合成され得る証拠を有する。最後に、公表された報告と一致して、本発明者らは、所与のリコンビナーゼでは、リコンビナーゼ補助鎖交換が効率的に起こるのに必要とされる最小のプライマー長があることを見出した。具体的には、27〜30塩基対より短いオリゴヌクレオチドは、uvsXには不充分な基質である。それでも、20〜27ヌクレオチド程度の短いプライマーは、ハイブリダイゼーション媒介性プライミングを補助するのに完全に充分であり得る。従って、長いプライマーと短い方のプライマーを組合せることにより、一方プライマーは侵入誘発性伸長に、他方はハイブリダイゼーション誘発性伸長に使用されるように、RPA反応を偏向させることができる。
規定の核酸配列の有無は、試料核酸と以前に性質が測定されている核酸プローブとでハイブリッドを形成させた後、かかる相互作用を検出する適切な方法を行なうことにより測定され得ることは、広く実証されている。例えば、DNA、RNAまたは他の主鎖オリゴヌクレオチドが空間的に分離され、支持体に固定化されているマイクロアレイは、広く使用されている。次いで、試料中の相同配列の有無が、適切なバッファーおよび固定化されとプローブと試料核酸間でのハイブリッド形成を可能にする温度条件下で、試料を共インキュベートすることにより測定される。この場合、試料およびプローブは、ともに、ハイブリダイズできるように完全または部分的に融解状態で提供される。標識が試料中に組み込まれている場合は、続いて、相互作用が定量され得る。
本発明者らは、低い一定の温度で核酸の大規模に幾何級数的な増幅を可能にすることを念頭に、動的なリコンビナーゼ活性に完璧な環境の確立に取り組んだ。しかしながら、確立された環境は、他の酵素との組合せで明白に使用され得、または実際にはそれらなしで、さまざまな状況において、一般的に古典的なハイブリダイゼーション反応と置き換え得る。例えば、分子的クローニング手順は、大きなトラクトのDNAを互いに、熱融解よりもずっとより有効に組換え、アニーリングする可能性を利用したものであり得る。また、インビトロ分子的プロセスに使用され得る多くの他の酵素は、大部分の中等温度好性の酵素と適合性の低温環境の恩恵を被り得る。かかる酵素としては、ポリメラーゼに加え、らせん変形(ゆがみ)認識ヌクレアーゼ(例えば、Slヌクレアーゼ)、FLAPエンドヌクレアーゼ、制限エンドヌクレアーゼ、塩基修飾または除去酵素、リアーゼ、ヘリカーゼ、トポイソメラーゼ、リガーゼ、逆転写酵素、RNase H活性、リゾルベース、RNAポリメラーゼ、および核酸に作用するか、または、これらと相互作用する任意の他の酵素が挙げられ得る。これらはまた、インビトロ系に適当なDNA代謝酵素に加え、検出プロトコルに使用される他の酵素、例えば、アルカリホスファターゼまたはホースラディッシュペルオキシダーゼを伴ない得る。安定で動的な組換え系特有の特性と他の触媒活性の組合せにより、潜在的に非常に多数の新たな方法および適用が可能になる。
RPAは非常に敏感な検出方法であるため、本発明者らは、他の超感受性の増幅プロトコル(例えば、PCRなど)で見られるキャリーオーバー混入物(莢雑物)の問題に直面した。本発明者らは、ここに、dUTPが、RPA反応において部分的または完全にdTTPの代用として使用され得、従って、先のRPA反応の産物を真の試料標的と識別する簡単な方法が提供されることを示す。RPA反応物をそれらの開始時にdUTP脱グリコシル化酵素を不活化する目的で熱処理すること(PCRプロトコルでは、混入物制御の場合に行なう)は望ましくないため、本発明者らは、脱グリコシル化酵素阻害剤を反応物と混合し、開始を可能にする代替アプローチを提案する(図61および62を参照)。
特異的RNA分子の存在の検出が所望される環境では、これを検出するためにRPA使用することが可能であるが、逆転写酵素活性を用いてRNAが、まずDNA形態に変換される。最も簡便なのは、逆転写およびRPAがすべての単一の均一な環境で行なわれる場合であろう。本発明者らは、逆転写RPA反応(RT−RPA)が単一のチューブ環境内で逆転写酵素を反応環境中に含め、他の若干の変形(ゆがみ)を伴なうことにより行なわれ得ることを示す(図60を参照)。
RPAの特徴は、携帯型易利用性診断用一体型製品の構成が理想的に適合される。ある一連の場合、特異的DNA増幅反応が起こったか否かの評価は、定量的解析の必要性が中程度にすぎない場合、2つの標識プライマーが物理的にアンプリコン内で会合しているか否かを評価する簡単な形式によって行なわれ得る。本発明者らは、ここに、この簡単な着想が、有効であり、特に、広く使用されている手法側方流動ストリップ系がこの役割を果たすのに理想的であることを示す。RPA反応物は、このような系のための試料ランバッファーと直接混合し得、アンプリコンの存在を、数分以内で測定し得る(図63を参照)。
本発明者らは、ここに、血液の単純なライセートが直接RPA反応において使用され得ることを示す。これは、RPA系を含む診断用産物の形式が、RPA反応物への直接添加前で、試料の処理ほんのわずかしか必要し得ない可能性を提供する(図64を参照)。
本発明者らは、ここに、RPAに使用される動的な組換え環境の存在により、PCRなどの他の手法における同等の環境と比べて、蛍光プローブの挙動を改変することを示す。最も特別には、フルオロフォアおよび消光剤を含有する二重標識プローブが使用され、オリゴヌクレオチド内で2つの基を分離している塩基の数はより少ないのがよく、おそらく、これは、飽和DNA結合タンパク質が、遊離した溶液中でB型二本鎖DNAとオリゴヌクレオチド存在するランダムコイルの両方の状態に対してプローブに伸長するためである(図73)。さらにまた、本発明者らは、かかるプローブとそれらの標的DNAとの二本鎖ハイブリッドを特異的にプロセッシングするために使用され得る重要な酵素を同定した。このようなアプローチは、リアルタイム「第3の」プローブストラテジーがRPAに関して構成されるはずであるアプローチ、およびPCRにおいて充分確立されたアプローチ、例えば、「Taqman」アプローチ、分子ビーコンなどとどのように違うのかを教示する。特に、本発明者らは、「Taqman」アプローチは、おそらく、大腸菌Pol Iなどの酵素と会合する5’ヌクレアーゼが、RPA反応を抑制するFLAPエンドヌクレアーゼ活性を有するため、RPAでは使用され得ないと決定した。
DNA配列は、図1に示すリコンビナーゼ−ポリメラーゼ増幅(RPA)法に従うリーディング鎖合成を用いて増幅され得る。図1は、RecA/プライマー負荷を示す。鋳型DNAおよび/またはポリメラーゼの添加前、RecAおよびSSBは、単鎖オリゴヌクレオチドプライマーへの結合に関して競合する。RecRおよびRecOの存在下では、RecAは、単鎖プライマー上に選択的に安定化され、RecOおよびRecRとの複合体状態のRecA核タンパク質フィラメントを形成する。この複合体は、二本鎖DNAを侵入し、オリゴヌクレオチドプライマー相同な部位にDループを形成する能力を有する。あるいはまた、RecA、RecOおよびRecRを、反応混合物へのSSBの導入前に、オリゴヌクレオチドプライマー上に前負荷し得る。
RPA反応は、実施例1に記載のようにして行なう。反応物の一部、10分の1(1/10)および100分の1(1/100)の一部を取り出し、第2のラウンドのRPAでのDNA鋳型の代わりに使用する。LsRPA、リーディング/ラギングRPA、およびその組合せが、ネステッドRPAに使用され得る。
DNA配列は、同時リーディング鎖/ラギング鎖合成を用い、図2に示すリコンビナーゼ−ポリメラーゼ増幅(RPA)法に従って、増幅され得る。この図は、lsRPAを具体的に示す。図2Aは、RecA/プライマー核タンパク質フィラメントが二本鎖鋳型DNAに侵入し、相同な標的部位と優先的に会合することを示す。Dループが形成され、合成が進行すると、置換された単鎖DNAは、SSBで被覆された状態になる(図2A)。二本鎖DNAからのRecA解放がATP加水分解によって、5’−3’方向に、またはヘリカーゼ/リゾルベースもしくはポリメラーゼ活性の結果として起こり得る(図2A)。合成が継続されるにつれて(図2B)、ポリメラーゼは、SSBが結合した被置換単鎖鋳型に出合う。二本鎖標的部位は、RecA/プライマー核タンパク質フィラメントによって再侵入される。後続のラウンドのlsRPAは、再侵入された部位から進行する(図2B)。
図18は、recA(C)をgp32(N)と、オリゴヌクレオチドであるTester3bio(5’ビオチン標識を有する)およびSizer(サイザー)1、Sizer2、Sizer3、Sizer4またはTester2の対の存在下で組合せた実験の結果を示す。これらの後者の非ビオチン化オリゴヌクレオチドは、共通のTester3bioオリゴヌクレオチドから進行的にさらに遠くなるように配置した。鋳型は、プラスミドから解放されるおよそ300bpの線状DNA断片であった。Tester3bioは、この断片の一方の末端に相補的となり、この配列に対して相対的な5’突出端を含むように設計した。
図19は、オリゴヌクレオチドであるTester3bio(5’ビオチン標識を有する)およびSizer2の対の存在下、実施例1で用いたものと同様の条件下で、recA(C)をgp32(N)と組合せた実験の結果を示す。PEG化合物またはPEG 1450は、表示したとおりに用い、0.5fモルの鋳型を出発鋳型量として用いた。この実施例では、鋳型の進行的な希釈を調べた。あるいはまた、本発明者らは、プライマーと重複する末端を有しない線状化出発鋳型(大腸菌ruvB遺伝子のClaI消化物を有するプラスミドを用いることにより)、およびクレノウ断片の希釈物の使用を探究した。正しいサイズの断片の増幅が、すべてのレーンで起こり、PEG化合物の存在下の0.5fモル出発鋳型の場合が最も強かった。
図24は、オリゴヌクレオチドであるTester3bioおよびSizer2の存在下で、uvsX(C)をgp32(N)と組合せた実験の結果を示す。この実験における鋳型DNAは、実施例1および2で使用した大腸菌ruvB遺伝子保有プラスミドのEcoRV消化物であった。Tester3bioは、およそ300塩基対断片の一方の末端を認識し、標的配列の末端に相対的な5’突出端を含んだ。Sizer2は、この鋳型の他方の鎖を認識した。このオリゴヌクレオチドは組み込まれた配列に指向され、その結果、その3’末端がTester3bioの末端から約3回半のらせん状回転となった。
図30は、数個のプライマー対を用いて特異的DNA断片をヒトゲノムDNAから増幅した実験の結果を示す。反応には、バクテリオファージT4 gp32(C)K3A、uvsX(C)およびuvsY(N)タンパク質、ならびにエキソヌクレアーゼ欠損クレノウ断片、ならびにADPおよびAMPを変換させるATP再生系を構成するタンパク質を含めた。特異的DNA断片を検出するため、本発明者らは、電気泳動により分離した反応産物をナイロン膜に移し、次いで、特有の非プライマー内部配列を認識するビオチン化プローブとハイブリダイズさせた。
図31は、数個のプライマー対を用いて特異的DNA断片をヒトゲノムDNAから増幅した実験の結果を示す。反応には、バクテリオファージT4 gp32(C)K3A、uvsX(C)およびuvsY(N)タンパク質、ならびにエキソヌクレアーゼ欠損クレノウ断片を含め、ADPおよびAMPを変換させるATP再生系を含めた。特異的DNA断片を検出するため、本発明者らは、電気泳動により分離した反応産物をナイロン膜に移した。次いで、特有の非プライマー内部配列を認識するビオチン化プローブとハイブリダイズさせた。
図32は、一対のプライマーを用いて特異的DNA断片をヒトゲノムDNAから増幅した実験の結果を示す。反応には、バクテリオファージT4 gp32(C)、uvsX(C)およびuvsY(N)タンパク質、ならびにエキソヌクレアーゼ欠損クレノウ断片、ならびにADPおよびAMPを変換させるATP再生系を構成するタンパク質を含めた。PEG 1450は、10%w/vで含めた。オリゴヌクレオチドの1つは、すべての反応産物が増幅の最後に観察され得るように5’−ビオチンを含んだ。試料を1時間目、2時間目および3時間目に採取し、どれだけ反応が進行したかを観察した。試料の1つでは、最小の量のuvsY(N)を用いた場合(50ng/μl)、正確な断片の増幅が観察された(レーン4の矢印を参照)。この断片はBstXIによって、予期されたサイズの断片に切断され、これは、主に二本鎖であったことを示す。しかしながら、断片は、反応中、同時に蓄積される見かけ上鋳型非依存性のバンドよりも存在度が低かった。このようなバンドのサイズおよび鋳型非依存性性質は、これらがプライマーアーティファクト、例えば、プライマー二量体および/またはスナップバック合成産物であることを示した。特異的断片の増幅の欠如は、50ng/μlより高いuvsY(N)濃度で、反応が最適以下で起こったことを示した。これは、後の実験で確認された。
図36は、一対のプライマーを用いて特異的DNA断片をヒトゲノムDNAから増幅した実験の結果を示す。反応には、バクテリオファージT4 gp32(C)、uvsX(C)およびuvsY(N)タンパク質、ならびにエキソヌクレアーゼ欠損クレノウ断片、またはBstポリメラーゼ、ならびにADPおよびAMPを変換させるATP再生系を構成するタンパク質を使用した。オリゴヌクレオチドの1つは、すべての反応産物が増幅の最後に観察され得るように5’−ビオチンを含んだ。増幅された断片を、反応物の少量の試料をアクリルアミドゲル上で泳動させることによりサイズによって断片を分離した後、可視化した。この実験では、非切断ヒトゲノムDNAを0コピー、45コピーから漸増させ、次いで、標的コピー数を2880まで倍増させた。この実験では、2つのポリメラーゼ種の各々について、バッファーおよび温度の両方に関してわずかに異なる条件を用いた。クレノウ断片を用いた反応を37℃で行ない、一方、Bstポリメラーゼを用いた反応は42℃で行なった。バッファー組成の詳細を図での説明に示す。
すべてのクローンを、大腸菌、T4ファージ、B.subtilisまたはPhi−29ファージからのPCR増幅産物のクローニングにより構築した。増幅に使用したすべてのストック生物は、DSMZの公の供給元から入手した。タンパク質発現に用いたクローン化したDNAは、一般的に、断片のPCR増幅中にNまたはC末端のいずれかにヘキサヒスチジンペプチドタグが挿入されるpETペクター(例えば、pET−21)にか、またはB.subtilis由来のPol I(Bsuポリメラーゼ)の場合はpQEペクター(例えば、pQE31)にクローン化した。この開示では、N末端タグを含有するすべてのタンパク質を(N)が続くタンパク質名、例えば、gp32(N)で示すか、またはタグをC末端に含有する場合は、(C)が続く名称、例えば、gp32(C)で示す。さらに、本発明者らは、他の場合では修飾タンパク質を作製するために、数個のクローンを構築した。これらとしては、天然タンパク質の最後の17アミノ酸残基の欠失を有するrecA(C)(recA(C)δl7で示す)が挙げられる。同様の形態のT4 UvsX(C)タンパク質を作製し、UvsX(C)δ21で示す。また、本発明者らは、リジン3またはアルギニン4のいずれかを修飾しているgp32の変異形態を構築した。
本発明者らは、数個の異なる標的DNAおよび多数のオリゴヌクレオチドをこの研究において使用した。鋳型の関連する部分の配列、およびオリゴヌクレオチドの配列を以下に示す。
RuvB遺伝子のΕcoRV断片の配列を以下に示す。
図9は、二本鎖DNA標的および標的化オリゴヌクレオチドの性質について調べた結果を示す。スーパーコイル鋳型または線状化DNAのいずれかを用いた実験は、recAが、ポリメラーゼ伸長を最も容易にスーパーコイルDNA上で、または線状化DNAの末端で補助できる中間体の形成を触媒することを示した。ビオチン化オリゴヌクレオチド、Tester3bioを、スーパーコイル標的DNA、またはEcoRVもしくはClaIで線状化した標的鋳型のいずれかとともにインキュベートした実験の結果を示す。これにより、それぞれオリゴヌクレオチドまたは組み込まれた配列と重複する末端が生成された。反応溶液は、20mM Tris−酢酸塩pH 7.9、10mM Mg−酢酸塩、13μg recA、1μg 大腸菌SSB、27mMクレアチンリン酸、1Uクレアチンキナーゼ、0.2μMTester3bio、3mM ATP、200μM dG、dCおよびdT;1mM dA、50Uクレノウ、0.5pモル鋳型、120ng recO、120ng recR、0.5μM dnaB、および0.5μM dnaC810を含んだ。大腸菌recOおよびrecRタンパク質、ならびにdnaBおよびdnaC810タンパク質をこの実験に含めたが、これらは、結果に有意に影響しなかった。37℃で2時間の反応後、反応物を沈殿させ、6%変性ゲル上で泳動させ、、ナイロン膜に移し、ストレプトアビジン−HRPとともにインキュベートした後、ECLを行ない、反応性物質を検出した。各反応において、0.5pモルの鋳型を用いた。ゲル上にサイズおよび量の対照として、0.5pモルのビオチン化PCR断片(標識CON)を含めた。他の反応成分および条件を図に示す。
クローンおよびタンパクは、上記の実施例11に記載のようにして作製した。
本発明者らは、この研究において、数個の異なる標的DNAおよび多数のオリゴヌクレオチドを使用した。オリゴヌクレオチドの配列を以下に示し、実験での鋳型標的を図39Bに示す。大腸菌RuvB遺伝子標的を線状ラン−オンアッセイに使用した(図39B)。この断片を含有する同量のプラスミド鋳型を、EcoRV(ほぼ300bp断片を放出する)または、ClaI(DNAを線状化する)のいずれかで切断した。等モル量の鋳型を、ラン−オン実験に使用した(20nMの各鋳型)。この鋳型のKpnI/ClaI断片の配列を以下に示す。EcoRV断片はこの配列内に組み込まれており、その部位を強調する。
本発明者らは、数個の供給源由来のヒトゲノムDNAを使用した。Promega製の混合集団の男性ゲノムDNAを利用した。また、男性個体試料由来のDNAを図38Aにおいて試験した。個体1および2は父と息子であった。個体2のDNAを、図39Eの実験に使用し、図39Fの実験についてのDNAは別の男性個体であった。ヒトおよびB.subtilisの配列の増幅に使用したオリゴヌクレオチドの配列は、以下の通りである:
図37は、RPA法の概略図を示す。図37A(i)では、リコンビナーゼタンパク質uvsXは、ATPの存在下で、単鎖オリゴヌクレオチドに協同的に結合する。核タンパク質フィラメントは、ATPをADPに活発に加水分解する。自発的な分解は、単鎖結合タンパク質gp32の競合的結合をもたらし得、uvsYタンパク質およびポリエチレングリコールによって、これは抑制され、再負荷が助長される。図37A(ii)では、相同なDNAが検出された場合、リコンビナーゼフィラメントが鎖交換を触媒する。図37A(iii)では、鎖交換により、検索鎖がその相補鎖とマッチングされ、鎖を置換し、次いで、gp32に結合される。リコンビナーゼが分解する。図37A(iv)では、ポリメラーゼが該構造に接近し、オリゴヌクレオチドを伸長させ、一層多くの元の鎖が置換される。図37B(i)では、2つの対向する標的化核タンパク質複合体が、それらのそれぞれの標的と再結合し、DNA合成が開始される。図37B(ii)では、ポリメラーゼ複合体が互いに出合い、ポリメラーゼの一方が解離する。図37B(iii)では、残ったポリメラーゼが合成を継続し、2つの親鎖を遊離させ、ポリメラーゼは、遊離3’末端に再結合し、したがって、両方の鎖の複製が起こる。図37B(iv)では、新たな標的化事象が起こる。第2のラウンドでは、標的化プライマーの一方が遊離末端を置換する。図37Cでは、DNA末端または組み込まれたDNA配列での鎖交換の産物の比較。
(A)オリゴヌクレオチドを示し、その上部に矢印があり、矢印の先端は、オリゴヌクレオチドの3’末端を示す。配列を作製したが、最も3’側の5個の塩基のうち4個は、パリンドロームを形成した。このことは実際面では充分回避され得る。(B)大部分の初期事象は、3’末端が一過的にその5’配列に折りたたまれ、一過的にワトソンクリック塩基対を形成することから構成されることが推測される。(C)偶発的に、ポリメラーゼは、成功裏に完全にこの構造を伸長させて、一端でターン(Hで示す)を有する長い二本鎖ヘアピンをもたらす。配列および元の逆相補鎖の5’−3’方向を、うすい矢印で示す。(D)ストラテジー1では、同一の配列の第2のリコンビナーゼ被覆されたオリゴヌクレオチドが、ヘアピンの相同な二本鎖本体を標的化し、したがって、これをほぐすと推測される。この部分への相補鎖が工程Cで正しく生成されなかったため、ミスマッチが、侵入オリゴヌクレオチドの3’領域で起こる。4つの塩基対のパリンドロームが、偶然に生成されなかった場合、ミスマッチは、ここに示した場合よりさらにより深刻になり得る。(E)ごくまれにポリメラーゼが、この中間体のミスマッチ3’領域を伸長し得、中央部にミスマッチを有する逆反復配列を生成させる。この構造はまた、2つの同一のプライマーが短い3’重複部を形成して、プライマー二量体の形成をもたらす場合の産物に類似し得る。このような構造は、容易に幾何学位相(geometric phase)増幅に入り得ない。(F)ストラテジー2では、(C)で形成された長いヘアピンが一端で少し開かれる。一部の報告により、recAがdsDNAに結合して短い二本鎖の融解を促進し得ることが示されているため、dsDNAへのリコンビナーゼの結合によって、この工程は増強され得る。(G)一過的に融解された3’末端(オリゴヌクレオチドの元の5’末端に逆相補的)が、(B)と同様の様式で折りたたまれ、伸長可能な構造を形成する。(H)ポリメラーゼ伸長により、大きなヘアピンが作られる。この場合、一方の末端は、親オリゴヌクレオチドと同一の二本鎖配列を含有する。(I)別の親オリゴヌクレオチドが、完全な二本鎖標的に侵入する。(J)(I)のポリメラーゼ伸長により、表示した構造を有する大きな逆反復配列が生成する。両方の末端は、完全なプライマー標的部位を含有し、そのため、この構造は、指数関数的に増幅し得る。興味深いことに、逆反復配列構造は、侵入および伸長が一方の末端から起こると、被置換鎖は急速に自身で折りたたまれ、(H)に示す構造を形成する傾向を示すことを意味する。この産物は、構造(J)となるために別の組換え事象を必要とし、したがって、より高度な組換え活性が、特にノイズ増幅に好都合であることが起こり得、低い組換え活性によって強く妨害される。
(A)実験に使用した6個のオリゴヌクレオチドの配列を示す。J1およびK2オリゴヌクレオチドは、B.subtilis SpoOB遺伝子座(配列番号:66および69 それぞれ)を標的化し、他に記載のオリゴヌクレオチドはこれらの誘導体であり、示されるように、シトシンまたはグアノシンのホモポリマーストレッチを保有する。このようなオリゴヌクレオチドは、対にしてインキュベートし、どのペアが最も効率的に標的を増幅するかを評価し得る。(B)6つのプライマーを単独またはすべての可能な組合せでインキュベートした結果を示す。反応物は、37℃で2時間インキュベートした。反応条件は、50mM Tris.Cl pH 8.4、2mM DTT、5%Carbowax 20M、80mM酢酸カリウム、200μM dNTPS、3mM ATP、20mMクレアチンリン酸、50ng/μl クレアチンキナーゼ、300nMの各関連するオリゴヌクレオチド、800ng/μl gp32、120ng/μl uvsX、25ng/μl uvsY、および28ng/μl Bsuポリメラーゼ、約1コピー/μl B.subtilisゲノムDNA(連続希釈により概算した)(20μl反応容量)であった。最適なプライマー対はJ1+K2であり、次にJ1C+K2Cであった。一般的に、ホモポリマーのシトシンストレッチは、核タンパク質フィラメントをより活性にし、グアノシンストレッチはあまり活性にしないようであった。
(A)単一の反応入れ子ストラテジーを概説する。活性な核タンパク質フィラメントの外側対は、DNAを急速に増幅するが、このようなプライマーはまた、かなりノイズ性であり得る。外側プライマー対は、比較的低く、ゲルで検出可能なレベルの産物が達成し得ないような充分低い濃度で維持され得る。内側オリゴヌクレオチドは低活性であるが、はるかにノイズが少ない。このような内側オリゴヌクレオチドは高レベルで維持される。活性は、本開示において詳述しているように、長さ、組成およびバックボーン特性を変更することにより調整され得る。外側プライマーは試料中の標的を、例えば100万倍の増幅により急速に富化する。低速だが明瞭な内側プライマーは、ノイズをほとんど/全く生じないが、感受性は低い。外側プライマーによる標的の富化は、適切な時間枠で低速の内側プライマーの作用による確固とした産物蓄積を可能にするのに充分である。(B)活性なプライマーと活性の低いプライマーとを組合せた増幅スキームを示す。活性なプライマーは、低い標的濃度でノイズに関与し得るが、低速オリゴヌクレオチドはアーティファクトに関与せず、そのため、プライマーの会合を反応の最後に試験した場合、標的が試料中に存在するか非存在であるかが明白に測定され得る。
(A)プライマー間の関係の概略的な表示。NEST3−28(GGATGAATAAGCTGCAGCTGATTAAAGG−配列番号:76)プライマーおよびNEST3−26(ATGAATAAGCTGCAGCTGATTAAAGG−配列番号:77)プライマー(これらは、プライマーJ1およびK2(それぞれの上記される配列番号:66および69)によって生成される増幅配列に相同である)の中間に配置されるPstI制限酵素部位を示す。(B)N3−26の5’−ビオチン化型の固定化の図示。これは、ストレプトアビジン被覆磁気粒子に結合させる。(C)捕捉実験のスキーム。オリゴヌクレオチドJ1およびK2を溶液相中で、ビーズ上に固定化したN3−26プライマーとともに合わせる増幅反応を確立する。反応物を90分間インキュベートし20分ごとに軽くたたいてビーズを分散させる。(これらは、5%PEG反応溶液中できわめてゆっくり沈降する)。インキュベーション期間の最後に、磁石を用いてビーズを集め、上清みを除去して解析する。ビーズを2回素早く水で洗浄し、次いで、30分間37℃で、過剰のPstI 酵素とともに適切なバッファー中でインキュベートする。再度、上清みを除去して解析する。
(A)プライマーApoB4およびApo300の配置(それぞれ、配列番号:20および21)、これらは、ヒトアポリポタンパク質B遺伝子座の断片と隣接する。(B)SYBR金は、RPA反応物中に、1:50,000以上での希釈度で、反応を完全に阻害することなく含め得る。反応物は、50mM Tris pH 8.4、80mM酢酸カリウム、200μM dATP、dCTP、dGTP、およびdTTP、50ng/μl クレアチンキナーゼ、1.5mM ATP、10mM酢酸マグネシウム、2mM DTT、30mM クレアチンリン酸、300nM ApoB4プライマー、300nM ApoB300プライマー、5%Carbowax 20M、360ng/μl gp32、86ng/μl uvsX、15ng/μl uvsY、35ng/μl Bsuポリメラーゼで構成した。表示した希釈度のSYBR金を反応物中に含めた。(C)RPA反応を、各々、最終容量50μlを有する96ウェルプレート内で確立した。標的DNAおよびSYBR金色素のみを欠く成分のマスターミックスを氷上で確立した。SYBR金の5つの異なる希釈物を試験し、ストックからの最終希釈度は、1:50,000、1:60,000、1:70,000、1:80,000、および1:100,000とした。各希釈について、2および20コピー/マイクロリットルの出発標的コピー密度を調べた。(TE中で予測されるコピー密度まで希釈したヒトゲノムDNA)。いったん反応物を氷上で充分混合し、プレートを、BIO−TEK FLX 800蛍光マイクロプレートリーダーの予備加温プレート(37℃)に移した。読み値を1分ごとに収集し、50μlの水で希釈した300ngのヒトゲノムDNAを装置の感度標準として使用した。データをMicrosoft Excelに転送し、各反応について、相対蛍光(任意)をインキュベーション時間に対してプロットした。(D)SYBR金ではなくSYBR緑をアッセイした以外は、(C)のようにして、実験を確立した。
非対称なプライマーRPA法を用いる標的DNAの増幅の一例として、標準的なRPA条件を、2つの違いを伴って用いる。第1に、一方のプライマーは、3’LNAキャッププライマーである。第2に、さらなるポリメラーゼ、φ29を使用する。これは、リコンビナーゼ中間体からの合成を開始することができない。
50mM Tris.Cl pH 7.85
10mM 酢酸マグネシウム
2mM ジチオトレイトール
80mM 酢酸カリウム
5% PEG化合物(Carbowax 20M)
15mM クレアチンリン酸
3mM ATP
200μM dNTP
12ng/μl クレアチンキナーゼ
0.3μM オリゴヌクレオチド1,3’LNAキャップ
0.3μM オリゴヌクレオチド2
350ng/μl T4 gp32
100ng/μl T4 uvsX
20ng/μl T4 uvsY
20ng/μl Bsuポリメラーゼ
20ng/μl φ29ポリメラーゼ(exo−, またはexo−減衰)
インキュベーションは33〜40℃で30分間〜2時間
実施例16:ケージ化(caged)ATPの脱保護を伴うリアルタイムRPAの開始
インプット鋳型DNAを定量化するためのATPパルス(pulsed)RPAの使用の一例として、反応を、いくつかの違いを伴う標準的なRPA反応として構築する。第1に、ATPの代わりにケージ化ATPを使用する。第2に、二本鎖DNA検出試薬、SYBR緑を使用してRPA産物を定量化する。ATPの各パルスで完全な合成が確保されるように、プライマーを、短い(<200bp)反応産物が生成されるように設計する。
50mM Tris.Cl pH 7.85
10mM 酢酸マグネシウム
2mM ジチオトレイトール
80mM 酢酸カリウム
5% PEG化合物(Carbowax 20M)
3mM ケージ化ATP
SYBR緑Iの市販のストック(Molecular Probes)の1:50,000希釈物
200μM dNTP
0.3μM オリゴヌクレオチド1
0.3μM オリゴヌクレオチド2
600ng/μl T4 gp32
150ng/μl T4 uvsX
25ng/μl T4 uvsY
20ng/μl Bsuポリメラーゼ
インキュベーションは33〜40℃
ATP脱ケージ化(uncaging)の各サイクルの前に、SYBR緑I蛍光(494nm 励起最大−521nm 発光最大)を試料中で測定する。検出された蛍光は定量化の基礎となる。1分ごとに、365nmの光の短いパルスをケージ化ATPに適用すると、リコンビナーゼ活性のバーストがもたらされる。各サイクルで測定した蛍光を、次いで、インプット鋳型標準と比較し、インプット試料鋳型の量が測定され得る。
50mM Tris.Cl pH 7.85
10mM 酢酸マグネシウム
2mM ジチオトレイトール
80mM 酢酸カリウム
5% PEG化合物(Carbowax 20M)
15mM クレアチンリン酸(構成に応じて含める)
200μM dNTP
* 3mM ケージ化ATP(構成に応じて含める)
12ng/μl クレアチンキナーゼ(構成に応じて含める)
0.3μM オリゴヌクレオチド1
0.3μM オリゴヌクレオチド2
350ng/μl T4 gp32
100ng/μl T4 uvsX
20ng/μl T4 uvsY
20ng/μl Bsuポリメラーゼ
次いで、反応を、ATPにより拡散させるか、または元の「内部拡散」ミックスに含まれるケージ化ATPを活性化することのいずれかにより開始する。ポロニーが、33〜40℃で30分間〜2時間でのインキュベーション後に生成される。
この実施例では、STRを、隣接するオリゴヌクレオチドプライマーによって増幅する。STRは、TPOXとして知られるマーカーであり、米国および欧州の両方で法医学的解析に使用される標準的なSTRマーカーセットにより一般的に使用されている。このマーカーは、5’−ACTGGCACAGAACAGGCACTTAGGGAACCC−3’(配列番号92)のプライマー1および5’−GGAGGAACTGGGAACCACACAGGTTAATTA−3’(配列番号93)のプライマー2の配列を有する隣接プライマーによって増幅される。このマーカーは、4ヌクレオチドAATGの反復を有し、この反復は、5〜14回の反復で変動する。
50mM Tris.Cl pH 7.85
10mM 酢酸マグネシウム
2mM ジチオトレイトール
80mM 酢酸カリウム
5% PEG化合物(Carbowax 20M)
15mM クレアチンリン酸
3mM ATP
200μM dNTP
12ng/μl クレアチンキナーゼ
0.3μM オリゴヌクレオチド1,5’−フルオレセイン−標識
0.05μM オリゴヌクレオチド2
600ng/μl T4 gp32
150ng/μl T4 uvsX
35ng/μl T4 uvsY
20ng/μl Bsuポリメラーゼ
インキュベーションは33〜40℃で30分間〜2時間
増幅反応では、プライマー不均衡によりある量の単鎖DNAが生成され、この単鎖DNAは蛍光標識される。増幅反応反応の最後に、反応混合物を、空間的に固定化された二本鎖プローブのアレイと直接接触させる。各プローブは、研究中のDNAの既知の多型性形態1つに対応し、この形態が試料中に存在すれば、作製され得るアンプリコンと同じ長さである。
50mM Tris.Cl pH 8.5
10mM 酢酸マグネシウム
10mM ジチオトレイトール
80mM 酢酸カリウム
5% PEG化合物(Carbowax 20M)
15mM クレアチンリン酸
3mM ATP
500μM dNTP
100ng/μl クレアチンキナーゼ
0.3μM MS2UP
0.3μM MS2DOWN
800ng/μl T4 gp32
120ng/μl T4 uvsX
30ng/μl T4 uvsY
70ng/μl Bsuポリメラーゼ
0.13 単位/μl RNaseインヒビター
MuMLV(RNase Hを含む)逆転写酵素 10単位/μl(Promega)
であった。
50mM Tris.Cl pH 8.5
10mM 酢酸マグネシウム
2mM ジチオトレイトール
100mM 酢酸カリウム
5% PEG化合物(Carbowax 20M)
15mM クレアチンリン酸
3mM ATP
200μM dNTP(dA, dC & dG)
100ng/μl クレアチンキナーゼ
0.3μM J1プライマー(B.subtilis DNA用)(配列番号:66)
0.3μM K2プライマー(B.subtilis DNA用)(配列番号:69)
630ng/μl T4 gp32
140ng/μl T4 uvsX
35ng/μl T4 uvsY
20ng/μl Bsuポリメラーゼ
100コピー/μl B.subtilisゲノムDNA
反応の産物は2%アガロースゲルで分離し、臭化エチジウムで染色した。左端のパネルに示したように、dUTPまたはdTTPを異なる反応で使用した。より詳しくは、上側の左端レーンは、200μM dTTPを含み、したがって、標準的なRPA反応の典型例であった。続く6つのレーンもまた、200μM dTTPを含んだが、この場合、dUTPもまた表示した濃度で存在した。下側左端パネルの最後の6つのレーンはRPA反応の産物を含み、これらはdTTPは含まず、表示した濃度(50〜800μM dUTP)のdUTPを使用した。すべての場合において、予測された断片の増幅が起こったが、顕著にdUTPの存在が、生成するアンプリコンの量を減少させ、多くの場合、スナップバック合成が起こったときに見られる「倍増(doubling−up)」現象を抑制した。
50mM Tris.Cl pH 8.5
10mM 酢酸マグネシウム
2mM ジチオトレイトール
80mM 酢酸カリウム
5% PEG化合物(Carbowax 20M)
25mM クレアチンリン酸
3mM ATP
200μM dNTP
100ng/μl クレアチンキナーゼ
0.3μM オリゴヌクレオチドApoB4(配列番号:20)
0.3μM オリゴヌクレオチドApoB300(配列番号:21)
420ng/μl T4 gp32
140ng/μl T4 uvsX
35ng/μl T4 uvsY
20ng/μl Bsuポリメラーゼ
(A)では、1μlの水または1μlの新鮮なヒト全血のいずれかを、RPA反応に添加した。産物を2%アガロースゲルで分離した。矢印は、予測されたアンプリコンの位置を示す。非特異的「プライマー」アーティファクトを、水対照および試料中に存在させた。おそらく、標的の非存在または非常に低コピーの標的の結果として起こる。本発明者らは、きわめて少ないコピーのゲノムDNAが血液試料の増幅での増幅に利用可能であると推測する。(B)では、lμlの血液をまず、10mM |Tris、1mM EDTA、120mM NaOH、0.1%SDS(表示のとおり)を含有する溶解溶液lμl、2μl、3μl、4μl、または5μlのいずれかと混合した試料を解析した。各場合で、lμlのそれぞれのライセートをRPA反応の開始に使用した。(したがって、1回の増幅で、より多くのバッファーで溶解したものは、より少ない血液試料を含んだ)。また、同じ実験での、水のみ(レーン1)または500コピーの標的(Promega)に相当する精製DNA(レーン7)を、増幅反応を開始するのに使用した試料も示す。4μlまたは5μlの溶解バッファーを使用した場合、赤血球の溶解は、明白に目視可能であり、溶液は粘性となった。本発明者らは、これらの試料がRPA反応の優れた開始物質になり、おそらく、大部分のDNAが接近可能な鋳型として放出されていることに注目する。
50mM Tris.Cl pH 8.5
10mM 酢酸マグネシウム
2mM ジチオトレイトール
80mM 酢酸カリウム(または、表示の通り)
5% PEG化合物(Carbowax 20M)(または、表示の通り)
25mM クレアチンリン酸
3mM ATP
200μM dNTP
100ng/μl クレアチンキナーゼ
0.3μM オリゴヌクレオチドJ1プライマー(配列番号:66)
0.3μM オリゴヌクレオチドK2(配列番号:69)
620ng/μl T4 gp32
140ng/μl T4 uvsX
35ng/μl T4 uvsY
SYBR緑(Invitrogen)のストックの1:50,000希釈物形態
20ng/μl Bsuポリメラーゼ。
反応の終了時に、反応物の一部を、スクロースローディングバッファーと混合し、そしてアガロースゲル上で分離し、そしてその後、臭化エチジウムで染色した。本発明者らは、高濃度の塩が反応を遅延するが、しかし十分に作用する広範な塩濃度があることに注目する。本発明者らは、4%から9%にPEG濃度を増大させることが、常に早い反応挙動を生じるが、しかし、ゲル系での産物の見掛けは、高いPEG濃度によって不利に影響されたことにも注目する。本発明者らは、高PGE濃度サンプルのスミアの多い外観が、非常に高い組換え活性によって発生されるDNAの進行中のネットワークから生じうることを推測する。これが、ゲル分析にとって望ましくないが、それは、蛍光を使用したリアルタイム分析にとって申し分なく適切でありうる。
リアルタイムRPA反応を、以下のとおりに確立した:
50mM Tris(pH8.5)
示されるとおりの6、10または16mM酢酸マグネシウム
2mM ジチオスレイトール
80mM 酢酸カリウム
5% PEG化合物(Carbowax−20M)
25mM クレアチンリン酸
3mM ATP
200μM dNTP
100ng/μl クレアチンキナーゼ
0.3μM オリゴヌクレオチドJ1プライマー(配列番号:66)
0.3μM オリゴヌクレオチドK2プライマー(配列番号:69)
600ng/μl T4gp32
120ng/μl T4uvsX
30ng/μl T4uvsY
20ng/μl Bsuポリメラーゼ
SYBR緑(Invitrogen)のストックからの1:50,000希釈物。
50mM Tris(pH8.4)
80mM 酢酸カリウム
10mM 酢酸マグネシウム
2mM DTT
5% PEG化合物(Carbowax−20M)
3mM ATP
20mM クレアチンリン酸
100ng/□l クレアチンキナーゼ
600ng/ml gp32
Apoプライマーについては200□M dNTP;CSFプライマーについては100□M dNTP
300n M各オリゴヌクレオチド。
50mM Tris(pH8.4)
80mM 酢酸カリウム
10mM 酢酸マグネシウム
2mM DTT
5% PEG化合物(Carbowax−20M)
3mM ATP
30mM クレアチンリン酸
100ng/μl クレアチンキナーゼ
420ng/μl gp32
140ng/μl UvsX
35ng/μl UvsY
200μM dNTP
300nMの各オリゴヌクレオチド
35ng/μl Bsuポリメラーゼ
25コピー/μl B.subtilisゲノムDNA
SYBR緑(Molecular probes)のストックから得た1:50,000希釈物
反応体積 50μl、37℃
であった。
50mM Tris(pH8.4)
80mM 酢酸カリウム
10mM 酢酸マグネシウム
2mM DTT
5% PEG化合物(Carbowax−20M)
3mM ATP
30mM クレアチンリン酸
100ng/μl クレアチンキナーゼ
420ng/μl gp32
140ng/μl UvsX
35ng/μl UvsY
200μM dNTP
300nMの各オリゴヌクレオチド
35ng/μl Bsuポリメラーゼ
20コピー/μl B.subtilisゲノムDNA
SYBR緑(Molecular probes)のストック由来の1:50,000希釈物
反応体積 50μl、37℃
であった。
50mM Tris(pH8.4)
80mM 酢酸カリウム
10mM 酢酸マグネシウム
2mM DTT
5% PEG化合物(Carbowax−20M)
3mM ATP
30mM クレアチンリン酸
100ng/μl クレアチンキナーゼ
200μM dNTP
示されるとおり120nMの各オリゴヌクレオチド
必要に応じて:
630ng/μl gp32
140ng/μl UvsX
35ng/μl UvsY
本発明者らは、オリゴヌクレオチドのみを含むFAMが、DNA結合タンパク質の存在下で僅かに消光され、同様に、フルオロフォアに密接に結合したタンパク質による蛍光のある程度の消光を示すことを観察する。しかし、明らかに対照的には、フルオロフォアと消光剤の両方を含有するオリゴヌクレオチドは、逆の現象を示し、そしてこれは、それらが、長さ10または15の残基であるかどうかによって、明らかに異なる。さらに特に、両方の後者のプローブは、タンパク質の不在下で明らかに消光され、ランダム・コイルの形成が、長さにかかわらず十分な消光を確保するという概念と一致する。しかし、タンパク質の存在下では、この消光は低減される。特に、15マーについての消光は、ほとんどなくなり、そしてそれは、タンパク質の存在下でオリゴヌクレオチドのみを含有するFAMの蛍光レベルに近いと想定する。逆に、10マーは、ほとんど影響されず、そして大きく消光されたままである。15マーに対する消光の減少は、DNA結合タンパク質の存在下で、プローブが、非常に堅いロッド(rod)に伸ばされるという示唆によって最も容易に説明される。例えば、UvsXおよびrecAは、単鎖DNAおよび二本鎖DNAを、等価なB形態のDNAの長さの約1.5倍に伸ばし、そして類似の伸長は、gp32について説明される。したがって、そして遊離溶液の挙動と対照的に、フルオロフォアと消光剤は、RPA環境で大きく分離され、DNA二本鎖ではよりいっそう分離され、さらにそしてハイブリダイゼーションでさらに消光される可能性がある(しかし、この実験では調査されない)。右側に、10マーおよび15マーのプローブが、リコンビナーゼ環境の不在下または存在下、および二本鎖DNAにハイブリダイズした場合に潜在的にどのようなの状態であるかを示す略図を供する。フルオロフォアと消光剤とをより大きな「励起」半径により示し、これらは重なったときに、十分な蛍光共鳴エネルギー移動、および蛍光発生の同時的な減少を導く。
50mM Tris(pH7.9)
80mM 酢酸カリウム
10mM 酢酸マグネシウム
2mM DTT
5% PEG化合物(Carbowax−20M)
3mM ATP
25mM クレアチンリン酸
100ng/μl クレアチンキナーゼ
600ng/μl gp32
120ng/μl UvsX
30ng/μl UvsY
100μM dNTP
200nMのJ1プライマー、K2プライマー、およびNEST26プライマー
100nMプローブ
30ng/μl Bsuポリメラーゼ
1000コピー/μl B.subtilisゲノムDNA、または示されるとおりの水対照。
50mM Tris(pH7.9)
80mM 酢酸カリウム
10mM 酢酸マグネシウム
2mM DTT
5% PEG化合物(Carbowax−20M)
3mM ATP
25mM クレアチンリン酸
100ng/μl クレアチンキナーゼ
600ng/μl gp32
120ng/μl UvsX
30ng/μl UvsY
100μM dNTP
200nMのJ1プライマー、200nMのK2またはL2プライマー、および200nM NEST26プライマー(レーン1−4);レーン5および6では、J1およびL2プライマーは、300nMであり、そしてNEST26を使用しなかった。
120nM プローブ
30ng/μl Bsuポリメラーゼ
1000コピー/μl B.subtilisゲノムDNA、または示される水対照
増幅反応物を、フェノール抽出、沈殿し、そして16.5%変性尿素−PAGEで分離した。分離した産物を、ナイロン膜に移動させ、そしてストレプトアビジン−HRP結合体を使用して産物を検出した。プローブの5’末端をビオチン化したので、このことは、未切断、切断および伸長プローブの検出を可能にした。(A)遊離、およびプロセッシングされたプローブの検出。標的断片を増幅し得るRPA環境の存在下で、そのプローブは、優先的に伸長されたことに注目する。明らかな伸長が、J1/K2プライマーの対について、標的が添加されない場合に起こる(混入によると思われる)一方で、J1/L2プライマー対が使用されるときに、これが起こらない(非常にかすかなバンドはサンプル交差ローディングのアーティファクトであると思われる)ことにも注目べきである。結局、本発明者らは、プローブ切断/伸長が、特異的な事象であることを推定する。さらに、鋳型J1/K2サンプルなしでの明らかな信号は、その系が、おそらく持ち込み混入から生じる、きわめてわずかな標的に対してでさえ応答性が高いことを示唆する。(C)では、J1とL2プライマーが、標的DNAなしか、またはマイクロリットル当たり1000コピーでの標的DNA(ゲノムDNA)を有するサンプルによりプローブと組合わせて使用された類似の実験の結果が示される。オリゴヌクレオチド濃度は以下の通りである:240nMでのJ1およびL2、ちょうど12nMでのプローブ、およびマイクロリットル当たり約1000ngのNfo酵素を使用した。他の条件は、パート(B)での実験について記述されるとおりである。サンプルを、所定の時間で取出し、そして停止した。サンプルを、フェノール抽出し、沈殿させ、そしてすでに示したとおり変性ゲルで泳動し、ナイロン膜に移動させ、そして上に記述されるとおり検出した。本発明者らは、切断/身長産物が、プローブが12nM程度に低い場合でさえ30分までに検出されうることに注目する。これは、これらの条件下でのNfo作用の反応速度が、リアルタイムで反応を良好にモニタリングするのに十分早いことを示唆する。
特定のDNA配列を増幅する、in vitro法に対する長く続く必要性が存在する。1980年代後半、ポリメラーゼ連鎖反応(PCR)法が、主にこの必要性を満たしてきた(R.K.Saiki et al.,Science 239,487−91(Jan 29,1988))。しかしながら、温度サイクル器具の必要性が、研究室の場面の外でのPCRの利用に対して、明らかな障壁となっている。本明細書で記述したように、本発明者らは、鋳型DNAの温度融解に対する必要性を取り除く、RPAと呼ぶ方法を開発した。RPAは、鋳型DNAに対するオリゴヌクレオチドプライマーのハイブリダイゼーションを媒介するための、決定的な規定条件下での、in vitroでバクテリオファージT4組換え/複製系の成分を組み合わせている。特に、バクテリオファージT4リコンビナーゼuvsX、単鎖DNA結合(SSB)タンパク質gp32、およびリコンビナーゼローディング因子uvsY、分子クラウディング剤とともに、忠実性の高い、in vitroリコンビナーゼ−媒介DNA標的化が可能になる。この標的化系を、大腸菌(E.coli)または枯草菌(B.subtilis)PolIクラスの酵素によって媒介された、鎖置換DNA合成と組み合わせた場合、有効な指数関数的DNA増幅が達成される。
Claims (28)
- 標的核酸分子のDNAを増幅するリコンビナーゼポリメラーゼ増幅方法であって、
該標的核酸分子は、第1のDNA鎖および第2のDNA鎖を含み、
該方法は、
(a)リコンビナーゼ因子を核酸プライマーと接触させてリコンビナーゼ因子/核酸プライマー複合体を形成する工程であって、該リコンビナーゼ因子/核酸プライマー複合体は単鎖領域をその3’末端に含む、工程;
(b)該リコンビナーゼ因子/核酸プライマー複合体を該標的核酸分子と接触させ、それにより該標的核酸分子の一部において二本鎖構造を形成する工程;
(c)該核酸プライマーの3’末端を、1種類以上のポリメラーゼとdNTPとを用いて伸長させて、二本鎖核酸と、核酸被置換鎖とを生成する工程;ならびに
(d)(b)および(c)を、所望の増幅度に達するまで反復することを介して、該反応を継続する工程
を含み、ここで、
該標的核酸分子の一部は該核酸プライマーの配列に対して相同な配列からなり、そして、該方法は、工程(b)および(c)において、1重量%〜12重量%または1容量%〜12容量%のポリエチレングリコール(PEG)の存在下において実施され、該1重量%〜12重量%または1容量%〜12容量%のPEGの存在が増幅を刺激する、方法。 - 前記PEGが、PEG1450、PEG8000、PEG10000、15,000〜20,000ダルトンの分子量を有するPEG化合物、ならびにそれらの組合せからなる群より選択される、請求項1に記載の方法。
- 前記工程(b)および(c)がアクセサリー因子の存在下で実施され、該アクセサリー因子が、単鎖結合タンパク質、ヘリカーゼ、リゾルベース、RuvA、RuvB、RuvC、RecG、PriA、PriB、PriC、DnaT、DnaB、DnaC、DnaG、DnaXクランプ負荷体、ポリメラーゼコア複合体、DNAリガーゼ、スライディングクランプおよびそれらの組み合わせからなる群より選択される、請求項1に記載の方法。
- 前記アクセサリー因子が、単鎖結合タンパク質、ヘリカーゼ、リゾルベースおよびそれらの組合せからなる群より選択される、請求項3に記載の方法。
- 前記アクセサリー因子が、単鎖結合タンパク質、RuvA、RuvB、RuvC、RecG、PriA、PriB、PriC、DnaT、DnaB、DnaC、DnaG、DnaXクランプ負荷体、ポリメラーゼコア複合体、DNAリガーゼ、スライディングクランプおよびそれらの組み合わせからなる群より選択される、請求項3に記載の方法。
- 前記核酸プライマーが、DNA、RNA、PNA、LNA、モルホリノ主鎖核酸、ホスホロチオレート主鎖核酸、およびそれらの組合せからなる群より選択される、請求項1に記載の方法。
- 前記方法がリコンビナーゼ因子/核酸プライマー複合体安定化因子の存在下で実施され、該リコンビナーゼ因子がRecAであり、そして、該核酸プライマーがssDNAである、請求項1に記載の方法。
- 前記安定化因子が、RecR、RecO、RecFおよびそれらの組合せからなる群より選択される、請求項7に記載の方法。
- 前記方法がgp32、大腸菌SSBタンパク質、T4 gp32タンパク質、およびgp32の誘導体からなる群より選択される単鎖安定化因子の存在下で実施され、該gp32の誘導体が、N末端にタグを有するgp32、C末端にタグを有するgp32、リシンではないアミノ酸による3位のリシンの置換を有するgp32、およびアルギニンではないアミノ酸による4位のアルギニンの置換を有するgp32、ならびにそれらの組み合わせからなる群より選択される、請求項1に記載の方法。
- 前記リシンではないアミノ酸がアラニンであり、そして、前記アルギニンではないアミノ酸がグルタミンまたはトレオニンである、請求項9に記載の方法。
- 前記方法がリコンビナーゼ負荷タンパク質の存在下で実施され、該リコンビナーゼ負荷タンパク質が、T4 uvsY、大腸菌recO、大腸菌recRならびにそれらの組合せからなる群より選択される、請求項1に記載の方法。
- 前記リコンビナーゼポリメラーゼ増幅方法が前記リコンビナーゼ因子の補因子をさらに含み、該補因子が、ATP、ATP−γ−S、ATP−β−S、ddATP、およびその組合せからなる群より選択される、請求項1に記載の方法。
- 前記リコンビナーゼポリメラーゼ増幅方法がADPをATPに変換するためのATP再生系をさらに含む、請求項1に記載の方法。
- 前記ATP再生系が、クレアチンリン酸およびクレアチンキナーゼを含む、請求項13に記載の方法。
- 前記リコンビナーゼポリメラーゼ増幅方法がピロリン酸塩をリン酸塩に変換するためのピロホスファターゼをさらに含む、請求項1に記載の方法。
- 前記1種類以上のDNAポリメラーゼが、原核生物のポリメラーゼ、真核生物のポリメラーゼおよびファージにコードされたポリメラーゼからなる群より選択される、請求項1に記載の方法。
- 前記真核生物のポリメラーゼが、pol−α、pol−β、pol−δ、pol−εならびにそれらの組合せからなる群より選択される、請求項16に記載の方法。
- 前記原核生物のポリメラーゼが、大腸菌DNAポリメラーゼIクレノウ断片、バクテリオファージT4 gp43 DNAポリメラーゼ、Bacillus stearothermophilus ポリメラーゼIラージフラグメント、φ−29 DNAポリメラーゼ、T7 DNAポリメラーゼ、Bacillus subtilis Pol Iラージフラグメント(Bsuポリメラーゼ)、大腸菌DNAポリメラーゼI、大腸菌DNAポリメラーゼII、大腸菌DNAポリメラーゼIII、大腸菌DNAポリメラーゼIV、大腸菌DNAポリメラーゼVならびにそれらの組合せからなる群より選択される、請求項16に記載の方法。
- 前記リコンビナーゼ因子が温度感受性であり、該温度感受性リコンビナーゼ因子は、許容温度で鎖侵入活性を有するが、非許容温度では鎖侵入活性を有さない、請求項1に記載の方法。
- ネステッドリコンビナーゼポリメラーゼ増幅方法であって、
(a)請求項1に記載のリコンビナーゼポリメラーゼ増幅方法を用い、第1のプライマーおよび第2のプライマーを用いてDNAの一領域を増幅し、第1の増幅産物を生成させる工程;
(b)第3のプライマーおよび第4のプライマーを用い、請求項1に記載のリコンビナーゼポリメラーゼ増幅方法を用いて該増幅産物を増幅し、第2の増幅産物を生成させる工程であって、該第2の増幅産物は、該第1の増幅産物内に含まれる、より短い配列である、工程;
を含む、方法。 - 前記dNTPのうちの少なくとも一部または少なくとも1つのプライマーが、検出可能なマーカーで標識されている、請求項1に記載の方法。
- 前記標識が、蛍光色素、酵素、蛍光消光剤、酵素阻害剤、放射性標識およびそれらの組合せからなる群より選択される、請求項21に記載の方法。
- 前記リコンビナーゼ因子が、uvsXまたはT4 uvsXである、請求項1に記載の方法。
- 請求項1に記載のDNAを増幅するリコンビナーゼポリメラーゼ増幅方法であって、
(a)以下の試薬
(1)少なくとも1種類のリコンビナーゼ;
(2)少なくとも1種類の単鎖DNA結合タンパク質;
(3)少なくとも1種類のDNAポリメラーゼ;
(4)dNTP、またはdNTPとddNTPとの混合物;
(5)PEG;
(6)バッファー;
(7)還元剤;
(8)ATP、ATP−γ−S、ATP−β−SまたはddATP;
(9)少なくとも1種類のリコンビナーゼ負荷タンパク質;
(10)第1のプライマー、および任意選択の第2のプライマー;ならびに
(11)標的核酸分子
を反応物中で合わせる工程;ならびに
(b)該反応物を所望の増幅度に達するまでインキュベートする工程
を含み、1重量%〜12重量%または1容量%〜12容量%の該PEGが増幅を刺激する、方法。 - 請求項1に記載のDNAを増幅するリコンビナーゼポリメラーゼ増幅方法であって、
(a)以下の試薬
(1)0.2〜12μMの濃度のuvsXリコンビナーゼ;
(2)1〜30μMの濃度のgp32単鎖DNA結合タンパク質;
(3)50〜5000単位/mlの濃度のT4 gp43 DNAポリメラーゼまたはBsuポリメラーゼ;
(4)1〜300μMの濃度のdNTP、またはdNTPとddNTPとの混合物;
(5)1重量%〜12重量%または1容量%〜12容量%の濃度のポリエチレングリコール;
(6)1mM〜60mMの濃度のTris−酢酸バッファー;
(7)1mM〜10mMの濃度のDTT;
(8)1mM〜10mMの濃度のATP;
(9)0.2μM〜8μMの濃度のuvsY;
(10)第1のプライマー、および任意選択の第2のプライマーであって、該第1のプライマーおよび第2のプライマーは50nM〜1μMの濃度である、プライマー;ならびに
(11)少なくとも1コピーの標的核酸分子
を反応物中で合わせる工程;
(b)該反応物を所望の増幅度に達するまでインキュベートする工程
を含み、
該ポリエチレングリコールが増幅を刺激する、方法。 - 請求項1に記載のDNAを増幅するリコンビナーゼポリメラーゼ増幅方法であって、
(a)以下の試薬
(1)100〜200ng/μl uvsXリコンビナーゼ;
(2)600ng/μl gp32;
(3)20ng/μlのBsuポリメラーゼまたはT4ポリメラーゼ;
(4)200μM dNTP;
(5)1mM DTT;
(6)3mMのATP、ATP−γ−S、ATP−β−SまたはddATP;
(7)16ng/μl〜60ng/μl uvsY;
(8)300nMの第1のプライマーおよび300nMの第2のプライマー;
(9)80mM 酢酸カリウム;
(10)10mM 酢酸マグネシウム;
(11)20mM クレアチンリン酸;
(12)100ng/μlクレアチンキナーゼ
を反応物中で合わせる工程;
(b)該工程(a)の試薬を凍結乾燥して凍結乾燥試薬を形成する工程;
(c)該凍結乾燥試薬を
(1)1mM〜60mMの濃度のTris−酢酸バッファー;
(2)1重量%〜12重量%または1容量%〜12容量%の濃度のポリエチレングリコール;
(3)標的核酸
により再構成する工程;
(d)該反応物を所望の増幅度に達するまでインキュベートする工程
を含み、
該ポリエチレングリコールが増幅を刺激する、方法。 - 請求項1に記載のDNAを増幅するリコンビナーゼポリメラーゼ増幅方法であって、
(a)以下の試薬
(1)100〜200ng/μl uvsXリコンビナーゼ;
(2)300〜1000ng/μl gp32;
(3)10〜50ng/μlのBsuポリメラーゼまたはT4ポリメラーゼ;
(4)50〜500μM dNTP;
(5)0.1〜10mM DTT;
(6)3mMのATP、ATP−γ−S、ATP−β−SまたはddATP;
(7)16ng/μl〜60ng/μl uvsY;
(8)50〜1000nMの第1のプライマーおよび50〜1000nMの第2のプライマー;
(9)40〜160mM 酢酸カリウム;
(10)5〜20mM 酢酸マグネシウム;
(11)10〜40mM クレアチンリン酸;
(12)50〜200ng/μl クレアチンキナーゼ;
を反応物中で合わせる工程;
(b)該工程(a)の試薬を凍結乾燥して凍結乾燥試薬を形成する工程;
(c)該凍結乾燥試薬を
(1)1mM〜60mMの濃度のTris−酢酸バッファー;
(2)1重量%〜12重量%または1容量%〜12容量%の濃度のポリエチレングリコール;
(3)標的核酸
により再構成する工程;
(d)該反応物を所望の増幅度に達するまでインキュベートする工程
を含み、
該ポリエチレングリコールが増幅を刺激する、方法。 - 請求項1に記載のDNAを増幅するリコンビナーゼポリメラーゼ増幅方法であって、
(a)以下の試薬
(1)100〜200ng/μl uvsXリコンビナーゼ;
(2)300〜1000ng/μl gp32;
(3)10〜50ng/μlのBsuポリメラーゼまたはT4ポリメラーゼ;
(4)50〜500μM dNTP;
(5)0.1〜10mM DTT;
(6)3mMのATP、ATP−γ−S、ATP−β−SまたはddATP;
(7)16ng/μl〜60ng/μl uvsY
(8)50〜1000nMの第1のプライマーおよび50〜1000nMの第2のプライマー;
(9)40〜160mM 酢酸カリウム;
(10)5〜20mM 酢酸マグネシウム;
(11)10〜40mM クレアチンリン酸;
(12)50〜200ng/μl クレアチンキナーゼ;
(13)1重量%〜12重量%または1容量%〜12容量%の濃度のポリエチレングリコール
を反応物中で合わせる工程;
(b)該工程(a)の試薬を凍結乾燥して凍結乾燥試薬を形成する工程;
(c)該凍結乾燥試薬を
(1)1mM〜60mMの濃度のTris−酢酸バッファー;
(2)標的核酸
により再構成する工程;
(d)該反応物を所望の増幅度に達するまでインキュベートする工程
を含み、
該ポリエチレングリコールが増幅を刺激する、方法。
Applications Claiming Priority (11)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US57616204P | 2004-06-01 | 2004-06-01 | |
| US57614804P | 2004-06-01 | 2004-06-01 | |
| US60/576,162 | 2004-06-01 | ||
| US60/576,148 | 2004-06-01 | ||
| US10/931,916 | 2004-09-01 | ||
| US10/931,916 US7399590B2 (en) | 2002-02-21 | 2004-09-01 | Recombinase polymerase amplification |
| US62229104P | 2004-10-26 | 2004-10-26 | |
| US60/622,291 | 2004-10-26 | ||
| US63274604P | 2004-12-02 | 2004-12-02 | |
| US60/632,746 | 2004-12-02 | ||
| PCT/IB2005/001560 WO2005118853A2 (en) | 2004-06-01 | 2005-04-11 | Recombinase polymerase amplification |
Related Child Applications (2)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2008096623A Division JP5089466B2 (ja) | 2004-06-01 | 2008-04-02 | リコンビナーゼポリメラーゼ増幅 |
| JP2011044524A Division JP2011103900A (ja) | 2004-06-01 | 2011-03-01 | リコンビナーゼポリメラーゼ増幅 |
Publications (3)
| Publication Number | Publication Date |
|---|---|
| JP2008500831A JP2008500831A (ja) | 2008-01-17 |
| JP2008500831A5 JP2008500831A5 (ja) | 2008-05-22 |
| JP5026958B2 true JP5026958B2 (ja) | 2012-09-19 |
Family
ID=35463460
Family Applications (4)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2007514201A Active JP5026958B2 (ja) | 2004-06-01 | 2005-04-11 | リコンビナーゼポリメラーゼ増幅 |
| JP2008096623A Active JP5089466B2 (ja) | 2004-06-01 | 2008-04-02 | リコンビナーゼポリメラーゼ増幅 |
| JP2011044524A Pending JP2011103900A (ja) | 2004-06-01 | 2011-03-01 | リコンビナーゼポリメラーゼ増幅 |
| JP2013029664A Active JP5856576B2 (ja) | 2004-06-01 | 2013-02-19 | リコンビナーゼポリメラーゼ増幅 |
Family Applications After (3)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2008096623A Active JP5089466B2 (ja) | 2004-06-01 | 2008-04-02 | リコンビナーゼポリメラーゼ増幅 |
| JP2011044524A Pending JP2011103900A (ja) | 2004-06-01 | 2011-03-01 | リコンビナーゼポリメラーゼ増幅 |
| JP2013029664A Active JP5856576B2 (ja) | 2004-06-01 | 2013-02-19 | リコンビナーゼポリメラーゼ増幅 |
Country Status (6)
| Country | Link |
|---|---|
| EP (4) | EP3540073B1 (ja) |
| JP (4) | JP5026958B2 (ja) |
| AU (1) | AU2005250233B2 (ja) |
| CA (1) | CA2569512C (ja) |
| ES (2) | ES2718482T3 (ja) |
| WO (1) | WO2005118853A2 (ja) |
Families Citing this family (71)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US7435562B2 (en) | 2000-07-21 | 2008-10-14 | Modular Genetics, Inc. | Modular vector systems |
| US7399590B2 (en) | 2002-02-21 | 2008-07-15 | Asm Scientific, Inc. | Recombinase polymerase amplification |
| US8030000B2 (en) | 2002-02-21 | 2011-10-04 | Alere San Diego, Inc. | Recombinase polymerase amplification |
| EP1992706B1 (en) * | 2002-02-21 | 2014-11-19 | Alere San Diego, Inc. | Recombinase polymerase amplification |
| JP2008501679A (ja) | 2004-06-02 | 2008-01-24 | エーエスエム サイエンティフィック, インコーポレイテッド | 2’−ニトロベンジル改変型リボヌクレオチド |
| WO2006053131A1 (en) | 2004-11-11 | 2006-05-18 | Modular Genetics, Inc. | Ladder assembly and system for generating diversity |
| ES2420831T3 (es) | 2005-07-25 | 2013-08-27 | Alere San Diego, Inc. | Procedimientos para multiplexar la ampliación de la recombinasa polimerasa |
| US8937181B2 (en) | 2006-04-13 | 2015-01-20 | Purdue Pharma L.P. | Benzenesulfonamide compounds and the use thereof |
| CA2650993C (en) * | 2006-05-04 | 2015-06-16 | Asm Scientific, Inc. | Recombinase polymerase amplification |
| AU2016204451B2 (en) * | 2006-05-04 | 2017-05-11 | Abbott Diagnostics Scarborough, Inc. | Recombinase Polymerase Amplification |
| JP4918409B2 (ja) | 2006-07-26 | 2012-04-18 | 西川ゴム工業株式会社 | 核酸配列の増幅方法 |
| DE102006054562A1 (de) * | 2006-11-20 | 2008-05-21 | Bayer Healthcare Ag | Verfahren zum Nachweis von Pyrophosphat mit Biolumineszenz Detektion |
| US9689031B2 (en) | 2007-07-14 | 2017-06-27 | Ionian Technologies, Inc. | Nicking and extension amplification reaction for the exponential amplification of nucleic acids |
| LT2660336T (lt) * | 2008-06-11 | 2017-10-10 | Orion Pharma (Uk) Limited | Izoterminė nukleorūgšties amplifkacija |
| WO2013158982A1 (en) | 2012-04-19 | 2013-10-24 | Life Technologies Corporation | Method of performing digital pcr |
| US9309557B2 (en) | 2010-12-17 | 2016-04-12 | Life Technologies Corporation | Nucleic acid amplification |
| US9334531B2 (en) | 2010-12-17 | 2016-05-10 | Life Technologies Corporation | Nucleic acid amplification |
| WO2010135310A1 (en) | 2009-05-20 | 2010-11-25 | Biosite Incorporated | Dna glycosylase/lyase and ap endonuclease substrates |
| EP2438196B1 (en) * | 2009-06-05 | 2016-12-21 | Alere San Diego, Inc. | Recombinase polymerase amplification reagents and kits |
| CN107739750A (zh) * | 2009-09-25 | 2018-02-27 | 艾利尔圣迭戈公司 | 粗基质中的核酸的检测 |
| KR101963462B1 (ko) | 2010-10-04 | 2019-03-28 | 제납시스 인크. | 재사용 가능한 자동화 평행 생물 반응 시스템 및 방법 |
| US9399217B2 (en) | 2010-10-04 | 2016-07-26 | Genapsys, Inc. | Chamber free nanoreactor system |
| US9184099B2 (en) | 2010-10-04 | 2015-11-10 | The Board Of Trustees Of The Leland Stanford Junior University | Biosensor devices, systems and methods therefor |
| EP3147374B1 (en) | 2010-12-17 | 2019-01-30 | Life Technologies Corporation | Methods for nucleic acid amplification |
| EP3978620A1 (en) * | 2011-04-07 | 2022-04-06 | Abbott Diagnostics Scarborough, Inc. | Monitoring recombinase polymerase amplification mixtures |
| US9926596B2 (en) | 2011-05-27 | 2018-03-27 | Genapsys, Inc. | Systems and methods for genetic and biological analysis |
| US9352312B2 (en) | 2011-09-23 | 2016-05-31 | Alere Switzerland Gmbh | System and apparatus for reactions |
| EP2785868B1 (en) | 2011-12-01 | 2017-04-12 | Genapsys Inc. | Systems and methods for high efficiency electronic sequencing and detection |
| JP5958034B2 (ja) * | 2012-04-11 | 2016-07-27 | 富士レビオ株式会社 | 分子ビーコン型プローブを用いた標的核酸の検出方法 |
| WO2014007289A1 (ja) * | 2012-07-05 | 2014-01-09 | 日本碍子株式会社 | 核酸クロマトグラフィー法、核酸クロマトグラフィー用組成物及びこれを含むキット |
| CN102816756B (zh) * | 2012-09-07 | 2014-04-16 | 江苏奇天基因生物科技有限公司 | 等温核酸扩增反应试剂及等温核酸扩增方法 |
| CN105051214B (zh) | 2013-03-15 | 2018-12-28 | 吉纳普赛斯股份有限公司 | 用于生物分析的系统和方法 |
| JP5904153B2 (ja) * | 2013-03-29 | 2016-04-13 | ソニー株式会社 | 核酸増幅反応用試料の調製方法、核酸増幅方法、固相状核酸増幅反応用試薬及びマイクロチップ |
| US9688979B2 (en) * | 2013-06-11 | 2017-06-27 | Biocartis Nv | Biomolecule drying process for long-term storage |
| EP3792921A1 (en) | 2013-12-11 | 2021-03-17 | Genapsys, Inc. | Systems and methods for biological analysis and computation |
| US9822401B2 (en) | 2014-04-18 | 2017-11-21 | Genapsys, Inc. | Methods and systems for nucleic acid amplification |
| EP2966177A1 (en) | 2014-07-09 | 2016-01-13 | Vetgenomics, S.L. | Methods for detecting target DNA sequences |
| CN104232622B (zh) | 2014-09-24 | 2016-09-14 | 中国人民解放军疾病预防控制所 | 一种用聚合酶螺旋反应恒温扩增核酸的方法及其应用 |
| EP3277837A1 (en) * | 2015-03-31 | 2018-02-07 | Qiagen GmbH | Efficiency improving ligation methods |
| GB201519565D0 (en) | 2015-11-05 | 2015-12-23 | Alere San Diego Inc | Sample preparation device |
| CN106011307A (zh) * | 2016-01-29 | 2016-10-12 | 中国动物卫生与流行病学中心 | H7亚型禽流感病毒核酸快速检测方法 |
| EP4155397B1 (en) * | 2016-02-10 | 2024-06-26 | The Regents of The University of Michigan | Detection of nucleic acids |
| CN105543402B (zh) * | 2016-03-02 | 2019-07-02 | 刘国宪 | 等温核酸扩增反应试剂及等温核酸扩增方法 |
| WO2017152122A2 (en) * | 2016-03-04 | 2017-09-08 | Alere San Diego Inc. | Automated nested recombinase polymerase amplification |
| US9617587B1 (en) | 2016-04-04 | 2017-04-11 | Nat Diagnostics, Inc. | Isothermal amplification components and processes |
| US11299777B2 (en) | 2016-04-04 | 2022-04-12 | Nat Diagnostics, Inc. | Isothermal amplification components and processes |
| DE102016108115B4 (de) | 2016-05-02 | 2019-05-09 | Agilent Technologies, Inc. - A Delaware Corporation - | Probenpuffer mit niedermolekularem Farbstoffstabilisator für Gelelektrophorese |
| GB201611469D0 (en) | 2016-06-30 | 2016-08-17 | Lumiradx Tech Ltd | Improvements in or relating to nucleic acid amplification processes |
| US10544456B2 (en) | 2016-07-20 | 2020-01-28 | Genapsys, Inc. | Systems and methods for nucleic acid sequencing |
| CN106399590B (zh) * | 2016-10-31 | 2019-12-13 | 中国人民解放军军事医学科学院微生物流行病研究所 | 呼吸道感染相关腺病毒的通用型核酸等温检测试剂 |
| WO2018160574A1 (en) * | 2017-02-28 | 2018-09-07 | Alere San Diego Inc. | Microfluidic devices and related methods |
| CA3076378A1 (en) | 2017-09-21 | 2019-03-28 | Genapsys, Inc. | Systems and methods for nucleic acid sequencing |
| CN108130369A (zh) * | 2017-12-29 | 2018-06-08 | 博迪泰(厦门)生物科技有限公司 | 一种检测乳牛短脊椎综合症的引物、试剂盒及检测方法 |
| GB2569965A (en) | 2018-01-04 | 2019-07-10 | Lumiradx Uk Ltd | Improvements in or relating to amplification of nucleic acids |
| JP7491576B2 (ja) | 2018-06-12 | 2024-05-28 | キージーン ナムローゼ フェンノートシャップ | 核酸増幅法 |
| DE102018222357A1 (de) * | 2018-12-19 | 2020-06-25 | Fraunhofer-Gesellschaft zur Förderung der angewandten Forschung e.V. | In-vitro-Verfahren zum Nachweis von mindestens einer Nukleinsäure, die sich bei einem Lebewesen im Vollblut außerhalb der Blutzellen befindet und Vorrichtung und Kit hierzu |
| EP4047091A4 (en) | 2019-10-18 | 2023-11-15 | Epigeneron, Inc. | METHOD FOR DETECTING A TARGET NUCLEIC ACID, METHOD FOR DETECTING A NUCLEIC ACID-BINDING MOLECULE AND METHOD FOR DETERMINING THE NUCLEIC ACID-BINDING CAPACITY |
| CN111197110A (zh) * | 2020-01-16 | 2020-05-26 | 浙江农林大学 | 用于检测猫泛白细胞减少症病毒的rpa-fld方法 |
| CN111518669A (zh) * | 2020-05-07 | 2020-08-11 | 安徽科技学院 | 一种核酸检测微流控芯片及其应用 |
| WO2021226754A1 (zh) * | 2020-05-09 | 2021-11-18 | 杭州梓晶生物有限公司 | 一种病毒重组酶-聚合酶扩增检测方法 |
| US20240279751A1 (en) | 2020-06-10 | 2024-08-22 | King Abdullah University Of Science And Technology | A rapid multiplex rpa based nanopore sequencing method for real-time detection and sequencing of multiple viral pathogens |
| CN111979303A (zh) * | 2020-08-11 | 2020-11-24 | 上海奕谱生物科技有限公司 | 一种核酸检测试剂盒、方法及其应用 |
| EP3985128A1 (en) | 2020-10-14 | 2022-04-20 | midge medical GmbH | Isothermal nucleic acid amplification methods for point-of-need diagnosis of emerging infectious diseases |
| US20230383338A1 (en) * | 2020-10-14 | 2023-11-30 | Midge Medical Gmbh | Isothermal nucleic acid amplification methods for point-of-need diagnosis |
| EP3995592A1 (en) * | 2020-11-09 | 2022-05-11 | midge medical GmbH | Multimeric isothermal nucleic acid amplification methods for point-of-need diagnosis |
| CN112501262A (zh) * | 2020-12-15 | 2021-03-16 | 苏州点晶生物科技有限公司 | 一种基于双重多酶介导的核酸扩增检测方法及试剂 |
| CN112708662B (zh) * | 2020-12-28 | 2021-08-31 | 阅尔基因技术(苏州)有限公司 | 抑制生物dna样本中非目标区域的核苷酸组合物及其应用 |
| EP4053293A1 (en) | 2021-03-03 | 2022-09-07 | midge medical GmbH | Protected isothermal nucleic acid amplification (pina) methods for point-of-need diagnosis of emerging infectious diseases |
| CN113981045A (zh) * | 2021-11-23 | 2022-01-28 | 广州达安基因股份有限公司 | 制备恒温扩增混合酶系的方法 |
| CN114262697B (zh) * | 2021-12-30 | 2022-12-13 | 南京巨匠生物科技有限公司 | 融合Bsu DNA聚合酶和Bsu DNA聚合酶突变体及其基因、质粒、基因工程菌 |
| CN115747187B (zh) * | 2022-12-09 | 2023-06-20 | 厦门康基生物科技有限公司 | 一种重组酶UvsX及其表达基因和应用 |
Family Cites Families (16)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US5223414A (en) | 1990-05-07 | 1993-06-29 | Sri International | Process for nucleic acid hybridization and amplification |
| DK0612352T3 (da) * | 1990-05-07 | 1997-06-30 | Daikin Ind Ltd | Diagnostiske anvendelser af dobbelt-D-sløjfedannelse |
| US5273881A (en) | 1990-05-07 | 1993-12-28 | Daikin Industries, Ltd. | Diagnostic applications of double D-loop formation |
| JP2739378B2 (ja) | 1990-11-09 | 1998-04-15 | アメリカ合衆国 | Dnaに標的を定める方法 |
| US6117679A (en) * | 1994-02-17 | 2000-09-12 | Maxygen, Inc. | Methods for generating polynucleotides having desired characteristics by iterative selection and recombination |
| US5705366A (en) * | 1994-09-15 | 1998-01-06 | Johnson & Johnson Clinical Diagnostics, Inc. | Coamplification of target nucleic acids using volume exclusion agent in reaction composition, test kit and test device useful therefor |
| US20040110167A1 (en) * | 1995-07-13 | 2004-06-10 | Gerdes John C. | Lateral flow system for nucleic acid detection |
| WO2001066688A1 (en) * | 2000-03-08 | 2001-09-13 | Imagene Technology, Inc. | Lateral flow pcr with amplicon concentration and detection |
| WO2002086167A1 (en) * | 2001-04-20 | 2002-10-31 | The Penn State Research Foundation | Methods for nucleic acid manipulation |
| AU2002341898A2 (en) * | 2001-09-28 | 2003-04-07 | University Of Delaware | Polymorphism detection and separation |
| US7244562B2 (en) * | 2001-11-01 | 2007-07-17 | Gene Check, Inc. | RecA assisted detection of mutations, single nucleotide polymorphisms and specific sequences |
| EP1992706B1 (en) * | 2002-02-21 | 2014-11-19 | Alere San Diego, Inc. | Recombinase polymerase amplification |
| JP2005532827A (ja) * | 2002-07-12 | 2005-11-04 | ブリティッシュ・バイオセル・インターナショナル・リミテッド | 側方フローアッセイ用のデバイスおよびその方法 |
| EP1539979B1 (en) * | 2002-09-20 | 2008-11-19 | New England Biolabs, Inc. | Helicase dependent amplification of nucleic acids |
| EP1403379A1 (en) * | 2002-09-24 | 2004-03-31 | QIAGEN GmbH | Enhanced coamplification of nucleic acids |
| JP2008018820A (ja) * | 2006-07-12 | 2008-01-31 | Nsk Ltd | 衝撃吸収式ステアリングコラム装置 |
-
2005
- 2005-04-11 ES ES10180482T patent/ES2718482T3/es active Active
- 2005-04-11 EP EP19153024.5A patent/EP3540073B1/en active Active
- 2005-04-11 WO PCT/IB2005/001560 patent/WO2005118853A2/en active Application Filing
- 2005-04-11 ES ES05741662T patent/ES2425354T3/es active Active
- 2005-04-11 AU AU2005250233A patent/AU2005250233B2/en active Active
- 2005-04-11 EP EP21192246.3A patent/EP3985125A1/en active Pending
- 2005-04-11 JP JP2007514201A patent/JP5026958B2/ja active Active
- 2005-04-11 EP EP10180482.1A patent/EP2336361B1/en active Active
- 2005-04-11 EP EP05741662.0A patent/EP1759012B1/en active Active
- 2005-04-11 CA CA2569512A patent/CA2569512C/en active Active
-
2008
- 2008-04-02 JP JP2008096623A patent/JP5089466B2/ja active Active
-
2011
- 2011-03-01 JP JP2011044524A patent/JP2011103900A/ja active Pending
-
2013
- 2013-02-19 JP JP2013029664A patent/JP5856576B2/ja active Active
Also Published As
| Publication number | Publication date |
|---|---|
| EP3985125A1 (en) | 2022-04-20 |
| EP1759012B1 (en) | 2013-05-22 |
| EP2336361A3 (en) | 2011-07-06 |
| WO2005118853A2 (en) | 2005-12-15 |
| AU2005250233B2 (en) | 2011-03-17 |
| EP2336361B1 (en) | 2019-01-23 |
| EP3540073A1 (en) | 2019-09-18 |
| WO2005118853A3 (en) | 2006-06-15 |
| JP5856576B2 (ja) | 2016-02-10 |
| ES2718482T3 (es) | 2019-07-02 |
| JP2008188020A (ja) | 2008-08-21 |
| EP3540073B1 (en) | 2021-08-25 |
| JP2011103900A (ja) | 2011-06-02 |
| JP2013102769A (ja) | 2013-05-30 |
| AU2005250233A1 (en) | 2005-12-15 |
| CA2569512C (en) | 2016-05-31 |
| JP2008500831A (ja) | 2008-01-17 |
| ES2425354T3 (es) | 2013-10-14 |
| EP1759012A2 (en) | 2007-03-07 |
| CA2569512A1 (en) | 2005-12-15 |
| JP5089466B2 (ja) | 2012-12-05 |
| EP2336361A2 (en) | 2011-06-22 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP5026958B2 (ja) | リコンビナーゼポリメラーゼ増幅 | |
| US10329603B2 (en) | Recombinase polymerase amplification | |
| US7666598B2 (en) | Recombinase polymerase amplification | |
| JP4340543B2 (ja) | リコンビナーゼポリメラーゼ増幅 | |
| AU2013201222B2 (en) | Recombinase Polymerase Amplification | |
| CA2921988C (en) | Recombinase polymerase amplification | |
| AU2015203225B2 (en) | Recombinase Polymerase Amplification | |
| Class et al. | Patent application title: Recombinase Polymerase Amplification Inventors: Olaf Piepenburg (Essex, GB) Colin H. Williams (Waltham, MA, US) Niall A. Armes (Suffolk, GB) Derek L. Stemple (Hertfordshire, GB) |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20080402 |
|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20080402 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20101202 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20110301 |
|
| A02 | Decision of refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A02 Effective date: 20111207 |
|
| A711 | Notification of change in applicant |
Free format text: JAPANESE INTERMEDIATE CODE: A712 Effective date: 20120328 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20120405 |
|
| A911 | Transfer to examiner for re-examination before appeal (zenchi) |
Free format text: JAPANESE INTERMEDIATE CODE: A911 Effective date: 20120530 |
|
| TRDD | Decision of grant or rejection written | ||
| A01 | Written decision to grant a patent or to grant a registration (utility model) |
Free format text: JAPANESE INTERMEDIATE CODE: A01 Effective date: 20120619 |
|
| A01 | Written decision to grant a patent or to grant a registration (utility model) |
Free format text: JAPANESE INTERMEDIATE CODE: A01 |
|
| A61 | First payment of annual fees (during grant procedure) |
Free format text: JAPANESE INTERMEDIATE CODE: A61 Effective date: 20120621 |
|
| FPAY | Renewal fee payment (event date is renewal date of database) |
Free format text: PAYMENT UNTIL: 20150629 Year of fee payment: 3 |
|
| R150 | Certificate of patent or registration of utility model |
Ref document number: 5026958 Country of ref document: JP Free format text: JAPANESE INTERMEDIATE CODE: R150 Free format text: JAPANESE INTERMEDIATE CODE: R150 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| RD04 | Notification of resignation of power of attorney |
Free format text: JAPANESE INTERMEDIATE CODE: R3D04 |
|
| RD02 | Notification of acceptance of power of attorney |
Free format text: JAPANESE INTERMEDIATE CODE: R3D02 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| S111 | Request for change of ownership or part of ownership |
Free format text: JAPANESE INTERMEDIATE CODE: R313113 |
|
| R350 | Written notification of registration of transfer |
Free format text: JAPANESE INTERMEDIATE CODE: R350 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |