-
Hintergrund der Erfindung
-
Gebiet der Erfindung
-
Die vorliegende Erfindung betrifft ein Verfahren zur Herstellung einer L-Aminosäure unter Verwendung eines Mikroorganismus.
-
Beschreibung des Standes der Technik
-
Als typische Verfahren zur Herstellung von Substanzen unter Verwendung von Mikroorganismen sind Verfahren zur Herstellung von L-Aminosäuren durch Fermentation bekannt. L-Aminosäuren werden nicht nur als Würzstoffe und Nahrungsmittel, sondern auch als Komponenten für verschiedene Nährmittelgemische für medizinische Zwecke eingesetzt. Außerdem werden sie als Additive für Tiernahrung, Reagenzien bei der Arzneimittelherstellung und in der chemischen Industrie und als Wachstumsfaktoren für die Produktion von L-Aminosäuren, wie L-Lysin und L-Homoserin, unter Verwendung eines Mikroorganismus eingesetzt. Als Mikroorganismen, die L-Aminosäuren durch Fermentation produzieren können, sind coryneforme Bakterien, Escherichia-Bakterien, Bacillus-Bakterien, Serratia-Bakterien und dergleichen bekannt.
-
Was die vorstehend beschriebene Produktion von Zielsubstanzen durch Fermentation betrifft, sind die meisten der Materialien, die als Ausgangsmaterialien eingesetzt werden, solche, die Saccharide enthalten, wie Restmelasse. Bei der Aminosäurefermentation oder der Nucleinsäurefermentation wird ebenfalls die Kultivierung unter Verwendung eines Saccharids als Ausgangsmaterial durchgeführt. Obwohl Zuckerrohr und dergleichen reichlich Stärke enthalten, wird es als solches kaum als Ausgangsmaterial eingesetzt, vielmehr wird es in den meisten Fällen als Zersetzungsprodukt verwendet, worin Stärke beispielsweise in Monosaccharide oder Disaccharide zersetzt ist. Bei dem Zersetzungsverfahren wird im Allgemeinen eine Lösung eines Zucker zersetzenden Enzyms, wie Amylase, verwendet, wodurch die Starke, die ein Polysaccharid ist, in relativ niedrigmolekulare Saccharide, wie Glucose, Maltose und Maltotriose, zersetzt wird.
-
Bei der Fermentation unter Verwendung von Gram-negativen Enterobakterien, wie Escherichia coli (E. coli) verursacht die Verwendung einer solchen Stärkezersetzungslösung ein Problem. Beispielsweise verbraucht E. coli die als Hauptkomponente vorhandene Glucose, E. coli leidet jedoch an der so genannten Glucoserepression, was bedeutet, dass Oligosaccharide, die zwei oder mehr Monosaccharide enthalten, wie Maltose, nur verbraucht werden, nachdem die Monosaccharide vollständig verbraucht worden sind. Wenn die Fermentation zu einem Zeitpunkt beendet wird, zu dem nur Glucose, welche die Hauptkomponente der Stärkezersetzungslösung ist, verbraucht worden ist, werden Oligosaccharide, wie Maltose, nicht assimiliert, sondern bleiben ungenutzt. Wenn beabsichtigt wird, Oligosaccharide nach dem Verbrauch der Glucose zu verbrauchen, muss außerdem die Kultivierungsdauer verlängert werden, was zu einer ineffektiven Verschwendung der Nutzungskosten führt.
-
Es ist bekannt, dass E. coli und Salmonella typhimurium im Allgemeinen an der Glucoserepression leiden. Das heißt, wenn beabsichtigt wird, Glucose zusammen mit anderen Kohlenstoffquellen, wie Lactose, Maltose und Glyzerin, zu assimilieren, wird zuerst Glucose assimiliert und später werden die anderen Kohlenstoffquellen assimiliert. Monod et al. haben entdeckt, dass wenn Lactose und Glucose als Kohlenstoffquellen eingesetzt wurden, ein Zweiphasenwachstum beobachtet wurde, d. h. eine so genannte Diauxie (Monod, J., Growth, 11, 223–247, 1947). Molekularbiologische Forschungen haben diesen Mechanismus aufgeklärt. Das heißt, IIAGlc (Glucose-PTS-Enzym II), welches als Phosphatdonor für Glucose in der Phosphatkaskade zum Zeitpunkt der Assimilation in dem Glucosephosphoenolatpyruvat-Zuckerphosphotransferasesystem, d. h. in dem so genannten PTS-System, wirkt, liegt in einem dephosphoryliertem Zustand vor. Die dephosphorylierte IIAGlc verursacht den so genannten Induktorausschluss, bei dem die dephosphorylierte IIAGlc die Aufnahme anderer Saccharide inhibiert (Postma P. W., Lengeler J. W. und Jacobson G. R.: in Escherichia coli and Salmonella: Cellular and Molecular Biology (Hrsg. Neidhardt F. C.), S. 1149–1174, 1996, ASM Press, Washington D. C.).
-
Die Aufnahme von Maltose in Escherichia coli leidet unter der Glucoserepression, die durch die Wechselwirkung zwischen der dephosphorylierten IIAGlc und dem MalK-Protein verursacht wird, welches das Aufnahmesystem für Maltose durch Nicht-PTS darstellt. Das heißt, wenn das Bakterium Glucose aufnimmt, ist IIAGlc in der Zelle im Überschuss vorhanden und bindet an das MalK-Protein, wobei die Aufnahme von Maltose inhibiert wird. Außerdem wurde ein mutierter Stamm, der verbesserte Aufnahme von Maltose in Anwesenheit eines Glucoseanalogons zeigte, erhalten, und es ist bekannt, dass dieser mutierter Stamm eine Mutation in dem malK-Gen hat, welches für das MalK-Protein codiert (Dean D. A. et al., Regulation of the Maltose Transport System of Escherichia coli by the Glucose-specific Enzyme III of the Phosphoenolpyruvate-Sugar Phosphotransferase System., J. Biol. Chem., 265 (34), 21005–21010, 1990; Kuhnau, S. et al., The Activities of the Escherichia coli MalK Protein in Maltose Transport and Regulation, and Inducer Exclusion Can 8e Separated by Mutations, J. Bacteriol., 173 (7), 2180–2186, 1991).
-
Außerdem wurde für IIAGlc ein mutierter Stamm berichtet, der ein mutiertes Protein enthält, welches eine verringerte Bindung mit Lactosepermease zeigt (Zeug, G. A. et al., Mutation alanalysis of the enzyme IIIGlc of the phosphoenolpyruvate phosphotransferease system in Escherichia coli. Res. Microbiol., 143, 251–261, 1992). Die Lactosepermease ist ein Enzym zur Aufnahme von Lactose, welche eines der Nicht-PTS-Saccharide ist.
-
Es ist jedoch nicht bekannt, ob die vorstehend genannten mutierten Stämme Maltose in Anwesenheit von Glucose gleichzeitig assimilieren.
-
Zusammenfassende Darstellung der Erfindung
-
Eine Aufgabe der vorliegenden Erfindung ist es, die Assimilationsfähigkeit eines Mikroorganismus für Oligosaccharide, insbesondere Maltose, bei der Produktion einer L-Aminosäure durch Fermentation unter Verwendung des Mikroorganismus mit einer Kohlenstoffquelle, die Glucose und Oligosaccharide, wie Starkezersetzungslösungen, enthält, zu verbessern.
-
Die Erfinder der vorliegenden Erfindung haben eingehende Studien durchgeführt, um die vorstehend genannte Aufgabe zu lösen. Als Ergebnis haben sie gefunden, dass ein Mikroorganismus, bei dem die Wechselwirkung zwischen dem IIAGlc-Protein von Glucose-PTS und einem Protein, das an der Nicht-PTS-Aufnahme von Maltose beteiligt ist, verringert oder ausgeschaltet wurde, Maltose selbst in Anwesenheit von Glucose assimilieren konnte, wodurch die vorliegende Erfindung gemacht wurde.
-
Das heißt, die vorliegende Erfindung stellt Folgendes bereit.
- (1) Ein Verfahren zur Herstellung einer L-Aminosäure unter Verwendung eines Mikroorganismus, welches das Kultivieren des Mikroorganismus in einem Medium unter Produktion und Anhäufung der L-Aminosäure in dem Medium und das Gewinnen der L-Aminosäure aus dem Medium umfasst,
wobei der Mikroorganismus ein mutanter oder rekombinanter Stamm eines Mikroorganismus ist, worin die Maltoseassimilation durch die Wechselwirkung zwischen dem IIAGlc-Protein von Glucose-PTS und dem MalK-Protein kontrolliert wird, die Wechselwirkung zwischen dem IIAGlc-Protein und dem MalK-Protein des mutanten oder rekombinanten Stammes verringert oder ausgeschaltet ist, der Stamm Glucose und Maltose aufnehmen kann und das Medium Glucose und Oligosaccharide als Kohlenstoffquellen enthält.
- (2) Das Verfahren nach (1), wobei das Oligosaccharid Maltose ist.
- (3) Das Verfahren nach (1) oder (2), wobei die Wechselwirkung zwischen dem IIAGlc-Protein von Glucose-PTS und MalK verringert oder ausgeschaltet ist, weil das in dem Mikroorganismus enthaltene MalK-Protein eine Mutation hat, die unter einer Mutation zum Substituieren eines Thr-Restes gegen den Ala-Rest an Position 124 und einer Mutation zum Substituieren eines Gln-Restes gegen den Leu-Rest an Position 327 ausgewählt ist.
- (4) Das Verfahren nach einem der Punkte (1) bis (3), wobei die Wechselwirkung zwischen dem IIAGlc-Protein von Glucose-PTS und MalK verringert oder ausgeschaltet ist, weil das in dem Mikroorganismus enthaltene IIIAGlc-Protein eine Mutation hat, die unter einer Mutation zum Substituieren eines Ser-Restes gegen den Gly-Rest an Position 47 und einer Mutation zum Substituieren eines Thr-Restes gegen den Ala-Rest an Position 76 ausgewählt ist.
- (5) Das Verfahren nach einem der Punkte (1) bis (4), wobei die Zielsubstanz aus der Gruppe ausgewählt ist, die aus L-Lysin, L-Threonin und L-Phenylalanin besteht.
- (6) Das Verfahren nach einem der Punkte (1) bis (5), wobei der Mikroorganismus ein Escherichia-Bakterium ist.
-
Erfindungsgemäß kann die Assimilationsfähigkeit eines Mikroorganismus für Oligosaccharide, insbesondere Maltose, bei der Herstellung einer L-Aminosäure durch Fermentation unter Verwendung des Mikroorganismus mit einer Kohlenstoffquelle, die Glucose und ein Oligosaccharid, beispielsweise eine Stärkezersetzungslösung, enthält, verbessert werden.
-
Kurze Erklärung der Zeichnungen
-
1 zeigt die Struktur des Plasmidvektors pTS1 mit einem temperaturempfindlichen Replikationsursprung.
-
2 zeigt das Wachstum von E. coli W3100(tyrA)malK1 in einem Medium, das Glucose und Maltose enthält.
-
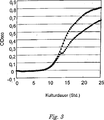
3 zeigt das Wachstum von E. coli W3100(tyrA)crr3 in einem Medium, das Glucose und Maltose enthält.
-
4 zeigt das Wachstum von E. coli W3100(tyrA)malK327 in einem Medium, das Glucose und Maltose enthält.
-
Bevorzugte Ausführungsformen der Erfindung
-
Im Folgenden wird die vorliegende Erfindung eingehend beschrieben.
-
Die Zielsubstanz, die durch den Mikroorganismus gemäß der vorliegenden Erfindung produziert wird, ist unter L-Aminosäuren, beispielsweise L-Threonin, L-Lysin, L-Glutaminsäure, L-Leucin, L-Isoleucin, L-Valin, L-Phenylalanin und dergleichen, ausgewählt. Besonders bevorzugte L-Aminosäuren sind L-Lysin, L-Threonin und L-Phenylalanin.
-
Der in der vorliegenden Erfindung eingesetzte Mikroorganismus ist ein Mikroorganismus, bei dem die Maltoseassimilation durch eine Wechselwirkung zwischen dem IIAGlc-Protein von Glucose-PTS und einem Protein, das bei der Nicht-PTS-Aufnahme von Maltose beteiligt ist, kontrolliert wird. Genauer gesagt können Bakterien der Gruppe der Enterobakterien genannt werden, wie Escherichia-Bakterien, Enterobacter-Bakterien, und Klebsiella-Bakterien, coryneforme Bakterien, Bacillus-Bakterien, Serratia-Bakterien und dergleichen. Es handelt sich vorzugsweise um einen Mikroorganismus, der Gensubstitutionen erlaubt. Ob ein Mikroorganismus für die vorliegende Erfindung eingesetzt werden kann oder nicht, kann beispielsweise durch Beobachten des Wachstums eines Wildtypstammes des Mikroorganismus in einem Medium, das Glucose und Maltose als Kohlenstoffquellen enthält, und durch Feststellen, ob das Zweiphasenwachstum, die so genannte Diauxie, beobachtet wird oder nicht, bestimmt werden. Wenn Diauxie beobachtet wird, wird angenommen, dass die Maltoseassimilation durch eine Wechselwirkung des IIAGlc-Proteins von Glucose-PTS und einem Protein, das an der Nicht-PTS-Aufnahme von Maltose beteiligt ist, kontrolliert wird.
-
Spezifische Beispiele von Mikroorganismen, die für die vorliegende Erfindung eingesetzt werden können, umfassen Escherichia coli AJ11442 (NRRL B-12185 und FERN BP-1543, vergleiche das
US-Patent Nr. 4,346,170 ), Brevibacterium lactofermentum AJ3990 (ATCC31269, vergleiche das
US-Patent Nr. 4,066,501 ) usw. für L-Lysin als Zielsubstanz, Escherichia coli VKPM B-3996 (RIA1867, vergleiche das
US-Patent Nr. 5,175,107 ), Corynebacterium acetoacidophilum AJ12318 (PERM BP-1172, vergleiche das
US-Patent Nr. 5,188,949 ) usw. für L-Threonin, Escherichia coli AJ12604 (FERN BP-3579, vergleiche die
Europäische Patentveröffentlichung Nr. 488,424 ), Brevibacterium lactofermentum AJ12637 (FERN BP-4160, vergleiche die
Französische Patentveröffentlichung Nr. 2,686,898 ) usw. für L-Phenylalanin, Escherichia coli AJ12624 (FERM BP-3853, vergleiche die
Französische Patentveröffentlichung Nr. 2,680,178 ), Brevibacterium lactofermentum AJ12475 (FERMBP-2922, vergleiche das
US-Patent Nr. 5,272,067 ) und dergleichen für L-Glutaminsäure, Escherichia coli AJ11478 (FERM P-5274, vergleiche die
Japanische Patentveröffentlichung (Kokoku) Nr. 62-34397 ), Brevibacterium lactofermentum AJ3718 (FERM P-2516, vergleiche das
US-Patent Nr. 3,970,519 ) usw. für L-Leucin, Escherichia coli KX141 (VKPM B-4781, vergleiche die
Europäische Patentveröffentlichung Nr. 519,113 ), Brevibacterium flavum AJ12149 (FERM BP-759, vergleiche das
US-Patent Nr. 4,656,135 ) usw. für L-Isoleucin, Escherichia coli VL1970 (VKPM B-4411, vergleiche die
Europäische Patentveröffentlichung Nr. 519,113 ), Brevibacterium lactofermentum AJ12341 (FERM BP-1763, vergleiche das
US-Patent Nr. 5,188,948 ) usw. für L-Valin und dergleichen.
-
Außerdem können, wenn die Zielsubstanz L-Lysin, L-Threonin oder L-Phenylalanin ist, Stämme, die durch Einführen eines Plasmids pVIC40, pCABD2 oder pMGAL1, die mit einem Gen, das bei der Produktion jeder der Aminosäuren beteiligt ist, in E. coli W3100 (tyrA) erhalten werden, auch in geeigneter Weise eingesetzt werden, wobei diese in den nachstehenden Beispielen beschrieben werden.
-
Außerdem kann in den Mikroorganismen, die für die vorliegende Erfindung eingesetzt werden, die Aktivität eines Proteins, das an der Produktion der Zielsubstanz beteiligt ist, verstärkt werden, oder die Aktivität eines Proteins, das an der Zersetzung oder dergleichen der Zielsubstanz beteiligt ist, kann in Abhängigkeit von der Zielsubstanz verringert werden.
-
Der für die vorliegende Erfindung eingesetzte Mikroorganismus ist ein mutierter oder rekombinanter Stamm, der aus einem vorstehend beschriebenen Mikroorganismus als Ausgangsstamm erhalten wird, wobei die Wechselwirkung zwischen dem IIAGlc-Protein von Glucose-PTS und einem Protein, das an der Nicht-PTS-Aufnahme von Maltose beteiligt ist, verringert oder eliminiert ist, es ist jedoch ein Mikroorganismus, der Glucose und Maltose aufnehmen kann. Das heißt, in der vorliegenden Erfindung enthalten das IIAGlc-Protein und das Protein, das an der Nicht-PTS-Aufnahme von Maltose beteiligt ist, eine Mutation, welche die Aufnahme von Glucose und Maltose im Wesentlichen nicht beeinflusst, obwohl sie die Wechselwirkung zwischen ihnen verringert oder ausschaltet.
-
In Escherichia coli wird das IIAGlc-Protein von dem crr-Gen codiert. Außerdem kann als Protein, das an der Nicht-PTS-Aufnahme von Maltose beteiligt ist, das MalK-Protein genannt werden, welches von dem malK-Gen in Escherichia coli codiert wird.
-
Um die Wechselwirkung des IIAGlc-Proteins und des Proteins, das an der Nicht-PTS-Aufnahme von Maltose beteiligt ist, zu verringern oder auszuschalten, kann eine Mutation, welche die Wechselwirkung zwischen diesen Proteinen verringert oder ausschaltet, in eines oder beide der Gene, die für diese Proteine codieren, eingeführt werden.
-
Ein crr-Gen oder malK-Gen, das eine vorstehend genannte Mutation aufweist, kann beispielsweise durch Isolieren des crr-Gens oder des malK-Gens aus einem mutierten Stamm, der in einem Medium, das Maltose als Kohlenstoffquelle und ein Glucoseanalogon, wie 2-Deoxyglucose, enthält, wachsen kann, erhalten werden. Als mutiertes malK-Gen, das wie vorstehend beschrieben erhalten werden kann, ist ein mutiertes malK-Gen mit einer Mutation zur Substitution eines Thr-Restes gegen den Ala-Rest 124 des codierten MalK-Proteins bekannt (Dean, D. A. et al., J. Biol. Chem., 265 (34), 21005–21010, 1990; Kuhnau, S. et al., J. Bacteriol., 173 (7), 2180–2186, 1991). Außerdem kann ein mutiertes malK-Gen, das für ein MalK-Protein codiert und eine Mutation aufweist, bei der ein Gln-Rest gegen den Leu-Rest in Position 327 ausgetauscht ist (L327Q-Typ Mutation), welche durch die Erfinder der vorliegenden Erfindung erhalten wurde, für die vorliegende Erfindung ebenfalls eingesetzt werden. Außerdem kann ein mutiertes malK-Gen, das für ein mutiertes MalK-Protein codiert, das sowohl eine Mutation, bei der ein Thr-Rest gegen den Ala-Rest in Position 124 ausgetauscht ist, als auch eine Mutation aufweist, bei der ein Gln-Rest gegen den Leu-Rest in Position 327 ausgetauscht ist, für die vorliegende Erfindung ebenfalls verwendet werden.
-
Andererseits ist ein Beispiel des mutierten crr-Gens ein crr-Gen, das eine Mutation, bei der ein Ser-Rest gegen den Gly-Rest in Position 47 des codierten IIAGlc-Proteins ausgetauscht ist, eine Mutation, bei der ein Thr-Rest gegen den Ala-Rest in Position 76 ausgetauscht ist, oder beide dieser Mutationen enthält.
-
Die Positionen der vorstehend genannten Mutationen werden von dem Met-Rest, welcher dem Initiationscodon entspricht, das als erstes Codon angenommen wird, nummeriert. Zusätzlich können das malK-Gen oder das crr-Gen eine oder mehrere Mutationen enthalten, welche keine erfindungsgemäßen Mutationen sind, sodass Deletionen, Substitutionen oder Insertionen eines oder mehrerer Aminosäurereste in dem codierten MalK-Protein oder IIAGlc-Protein auftreten können. Es können selbst solche malK-Gene oder crr-Gene für die vorliegende Erfindung eingesetzt werden, solange die Wechselwirkung zwischen dem MalK-Protein und dem IIAGlc-Protein verringert oder ausgeschaltet ist und die Aufnahme von Glucose und Maltose im Wesentlichen nicht beeinträchtigt wird. Wenn das MalK-Protein oder das IIAGlc-Protein eine Deletion oder Insertion eines oder mehrerer Aminosäurereste aufweist, sollten sich die Positionen der vorstehend genannten Mutationen verändern. Wenn beispielsweise das MalK-Protein eine Deletion eines Aminosäurerestes auf der N-terminalen Seite des Leu-Restes in Position 327 aufweist, sollte der Leu-Rest 327 die Position 326 bekommen. Auch in einem solchen Fall entspricht der Leu-Rest in Position 326 dem Leu-Rest in Position 327 eines Wildtypproteins. Deshalb sollen in der vorliegenden Beschreibung die Positionen der Mutationen Positionen darstellen, welche den Positionen in einem Wildtypgen oder einem Wildtypprotein entsprechen.
-
Als Verfahren zum Einführen der vorstehend genannten Mutationen in das malK-Gen und/oder das crr-Gen kann ein Verfahren zum Einführen einer Zielmutation in das malK-Gen und/oder crr-Gen durch ortsspezifische Mutagenese oder dergleichen, wobei ein mutiertes Gen erhalten wird, und das Substituieren des erhaltenen mutierten Gens gegen das malK-Gen und/oder crr-Gen auf einem Chromosom des Mikroorganismus durch Gensubstitution unter Einsatz der homologen Rekombination genannt werden.
-
Eine solche Gensubstitution kann beispielsweise auf dieselbe Weise wie die Gensubstitution unter Verwendung des nachstehend beschriebenen temperaturempfindlichen Plasmids durchgeführt werden. Beispiele der temperatursensitiven Plasmide von Escherichia coli umfassen pMAN031 (J. Bacteriol., 162, 1196, 1985), pMAN997 (
WO 99/03988 ) und dergleichen. Diese Plasmide können in Escherichia coli zumindest bei 30°C autonom replizieren, sie können jedoch bei 37–42°C nicht autonom replizieren.
-
Außerdem kann ein mutierter Stamm mit einer Zielmutation in dem malK-Gen und/oder crr-Gen auch durch Behandeln eines Mikroorganismus durch UV-Bestrahlung oder mit einem Mutagenisierungsmittel, das für eine übliche Mutagenisierungsbehandlung eingesetzt wird, wie N-Methyl-N'-nitro-N-nitrosoguanidin (NTG) oder salpetrige Säure, und Selektieren eines Stammes, der in einem Medium wachsen kann, welches ein Glucoseanalogon, wie 2-Deoxyglucose, enthält, erhalten werden.
-
Ob ein erhaltener Kandidatenstamm ein Zielmutantenstamm ist oder nicht, kann durch Isolieren des malK-Gens oder des crr-Gens aus dem Kandidatenstamm und Untersuchen seiner Nucleotidsequenz um die Mutationsstelle herum bestätigt werden.
-
Als Medium, das für die Kultivierung des erfindungsgemäßen Stammes eingesetzt wird, können herkömmlicherweise eingesetzte gut bekannte Medien in Abhängigkeit von der Art des zu verwendenden Mikroorganismus eingesetzt werden. Das heißt, übliche Medien, die eine Kohlenstoffquelle, Stickstoffquelle, anorganische Ionen und andere organische Komponenten, je nach Bedarf, enthalten, können eingesetzt werden. Es ist jedoch bevorzugt, ein Medium einzusetzen, das Glucose und ein Oligosaccharid, wie Maltose, als Kohlenstoffquellen enthält.
-
Als Kohlenstoffquelle, die nicht Glucose und Maltose ist, können Zucker, wie Lactose, Galactose und Stärkehydrolysat, Alkohole, wie Glycerin und Sorbitol, organische Säuren, wie Fumarsäure, Citronensäure und Bernsteinsäure, und dergleichen eingesetzt werden.
-
Als Stickstoffquelle können anorganische Ammoniumsalze, wie Ammoniumsulfat, Ammoniumchlorid und Ammoniumphosphat, organischer Stickstoff, wie Sojabohnenhydrolysat, Ammoniakgas, wässriges Ammoniak und dergleichen eingesetzt werden.
-
Als organische Spurennährstoffquellen können geeignete Mengen der erforderlichen Substanzen, wie Vitamin B1, L-Homoserin und L-Tyrosin, Hefeextrakt und dergleichen vorzugsweise enthalten sein. Zusätzlich zu diesen werden nach Bedarf kleine Mengen von Kaliumphosphat, Magnesiumsulfat, Eisenionen, Manganionen und dergleichen zugegeben.
-
Die Kultivierung kann unter bekannten Bedingungen durchgeführt werden, die herkömmlicherweise in Abhängigkeit von dem einzusetzenden Mikroorganismus eingesetzt werden. Beispielsweise wird die Kultivierung vorzugsweise unter aeroben Bedingungen während 16–120 Stunden durchgeführt. Die Kultivierungstemperatur wird auf 25–45°C kontrolliert, und der pH-Wert wird während der Kultivierung auf 5–8 kontrolliert. Anorganische oder organische saure oder alkalische Substanzen sowie Ammoniakgas und dergleichen können für die pH-Werteinstellung verwendet werden.
-
Um eine L-Aminosäure aus einem Medium nach Beendigung der Kultivierung zu gewinnen ist erfindungsgemäß kein spezielles Verfahren erforderlich. Das heißt, die Gewinnung einer L-Aminosäure kann durch eine Kombination bekannter Verfahren durchgeführt werden, beispielsweise solche unter Verwendung eines Ionenaustauscherharzes, Ausfällung und dergleichen.
-
Beste Ausführungsform der Erfindung
-
Im Folgenden wird die vorliegende Erfindung anhand der folgenden Beispiele genauer erklärt. Die in den folgenden Beispielen verwendeten Reagenzien wurden von Wako Pure Chemicals oder Nakarai Tesque bezogen, wenn nichts anderes angegeben ist. Die Zusammensetzungen der in jedem Beispiel verwendeten Medien sind nachstehend gezeigt. [L-Medium]
| Bactotryptonpepton (DIFCO) | 10 g/l |
| Hefeextrakt (DIFCO) | 5 g/l |
| NaCl | 5 g/l |
-
Es wurde 20 Minuten bei 120°C autoklaviert. [L-Agarmedium]
| L-Medium | |
| Bactoagar (DIFCO) | 15 g/l |
-
Es wurde 20 Minuten bei 120°C Dampf sterilisiert. [SOC-Medium]
| Bactotryptonpepton (DIFCO) | 20 g/l |
| Hefeextrakt (DIFCO) | 5 g/l |
10 mM NaCl
2,5 mM KCl
10 mM MgSO
4 10 mM MgCl
2 20 mM Glucose
-
Die Komponenten, ausgenommen die Magnesiumlösung und Glucose, wurden autoklaviert (120°C, 20 Minuten), dann mit 2 M Magnesiumstammlösung (1 M MgSO
4, 1 M MgCl
2) und 2 M Glucoselösung versetzt, wobei diese Lösungen vorher durch einen 0,22 μm Filter und dann nochmals durch einen 0,22 μm Filter filtriert worden waren. [M9-Minimalmedium]
| Na2HPO4·12H2O | 80 g/l |
| KH2PO4 | 15 g/l |
| NaCl | 2,5 g/l |
| NH4Cl | 5 g/l |
| MgSO4·7H2O | 246,48 mg/l |
| Saccharid (Glucose oder Maltose | |
| oder Gemisch dieser beiden bei | |
| einem geeigneten Verhältnis) | 5 g/l |
| pH 7,0 | |
-
MgSO
4 und Glucose wurden getrennt voneinander sterilisiert (120°C, 20 Minuten) und zugegeben. Eine geeignete Menge an Aminosäuren und Vitaminen wurden nach Bedarf zugegeben. Der pH-Wert wurde mit NaOH eingestellt. [M9-Minimalagarmedium]
| M9-Minimalmedium | |
| Bactoagar (DIFCO) | 15 g/l |
[Aminosäureproduktionsmedium]
| (NH4) 2SO4 | 20 g/l |
| KH2PO4 | 1 g/l |
| MgSO4·7H2O | 1 g/l |
| FeSO4·7H2O | 10 g/l |
| MnSO4·4H2O | 10 g/l |
| Hefeextrakt (DIFCO) | 2 g/l |
| Saccharid (Glucose oder Maltose | |
| oder Gemisch dieser beiden bei | |
| einem geeigneten Verhältnis) | 40 g/l |
| L-Tyrosin | 100 g/l |
| CaCO3 (Japanisches Arzneibuch) | 30 g/l |
| Streptomycin | 50 mg/l |
-
Das Saccharid, MgSO4·7H2O und Streptomycin wurden getrennt voneinander sterilisiert. Die anderen Komponenten wurden gemischt, mit KOH auf einen pH-Wert von 7,0 eingesteht und 10 Minuten bei 115°C autoklaviert. CaCO3 wurde 2 Tage bei 180°C einer Trockensterilisation unterzogen. Streptomycin wurde durch Filtration sterilisiert.
-
Beispiel 1: Einführung einer Mutation in das malK-Gen und Bestätigung der Verbesserung der Maltoseassimilationseigenschaft
-
Eine Kolonie von E. coli W3100 wurde in 5 ml L-Medium eingeimpft und über Nacht unter Schütteln kultiviert. Aus den erhaltenen Zellen wurde die chromosomale DNA unter Einsatz eines Wizard Genomic DNA Purification Kit (Promega) präpariert. Die PCR wurde unter Verwendung der vorstehenden chromosomalen DNA als Matrize und den nachstehend gezeigten Primern durchgeführt.
-
-
-
Die PCR wurde unter Verwendung von Pyrobest DNA-Polymerase, hergestellt von Takara Shuzo, und gemäß der dem Enzym beigefügten Anweisung durchgeführt. Nach dem vollständigen Ablauf der Reaktion wurde das Amplifikationsprodukt unter Verwendung eines BKL-Kit, hergestellt von Takara Shuzo, abgestumpft und phosphoryliert. Das amplifizierte Fragment wurde unter Verwendung des Ligation Kit ver.2 (Takara Shuzo) an pSTV28 (Takara Shuzo), der mit einem Restriktionsenzym Sma I (Takara Shuzo) und dann dephosphoryliert worden war, ligiert. Dieses Ligationsreaktionsgemisch wurde nach dem Verfahren von Hanahan et al. in E. coli JM109 transformiert (Hanahan, D., Studies an transformation of Escherichia coli with plasmids, J. Mol. Biol., 166, 557–580, 1983). Die Auswahl der Transformanten wurde auf einem L-Agarmedium durchgeführt, welches 50 μg/ml Chloramphenicol (Cm), 0,2 mM IPTG (Isopropyl-1-thio-β-D-galactopyranosid) und 40 μg/ml X-gal (5-Brom-4-chlor-3-indolyl-β-D-galactopyranosid) enthielt. Aus den Transformanten wurde auf herkömmliche Weise ein Plasmid extrahiert, und die Nucleotidsequenz des eingefügten Fragments wurde bestimmt, um zu bestätigen, dass das malK-Gen in die Smal-Schnittstelle von pSTV28 eingeführt worden war. Das Plasmid wurde pSTVmalK genannt.
-
Die Nucleotidsubstitution des malK-Gens auf pSTVmalK wurde wie folgt durchgeführt. Es wurde beschlossen, die Substitution von A gegen G in der Position 370 (Substitution von Thr gegen den Ala-Rest in Position 124 des MalK-Proteins) einzuführen. Die Positionen in den Nucleotidsequenzen, die hier verwendet werden, werden von dem A des Initiationscodons ATG an nummeriert, welches als das erste Nucleotid angenommen wurde, und die Positionen der Aminosäurereste werden von dem Met-Rest, welcher dem Initiationscodon entspricht und als die erste Aminosäure angenommen wurde, nummeriert.
-
Zunächst wurde die Substitution des Nucleotids auf dem Plasmid unter Verwendung von QuickChangeTM Site-Directed Mutagenesis Kit (STRATAGENE) durchgeführt. Die Sequenzen der für das Einführen der malK-Mutation eingesetzten Primer sind wie folgt.
-
-
-
Die Einführung der Mutation wurde durch Bestimmung der Nucleotidsequenz der betroffenen Stelle gemäß der dem Kit beigefügten Anweisung bestätigt. Das produzierte Plasmid wurde pSTVmalK-A124T genannt. Das Plasmid wurde mit den Restriktionsenzymen EcoR I und Hind III (beide von Takara Shuzo) verdaut und an dieselben Restriktionsenzymschnittstellen eines Plasmidvektors pTS1 mit einem temperaturempfindlichen Replikationsursprung ligiert.
-
pTS1 wurde durch Austauschen der Pst-I-Hind-III-Fragmente von pMAN031 (Matsuyama, S. und Mizushima S., Construction and characterization of a deletion mutation lacking micF, a proposed regulatory gene for OmpF synthesis in Escherichia coli., J. Bacteriol., 162 (3), 1196–1202, 1985) und pBR322 (von Takara Shuzo) erhalten (1). Das hergestellte Plasmid wurde pTSmalK-A124T genannt.
-
Die homologe Rekombination des malK-Gens mit malK auf dem Chromosom von E. coli W3100 (tyrA) (vergleiche die
Europäische Patentveröffentlichung Nr. 488,424 ) gemäß einem üblichen Verfahren für die homologe Rekombination (Matsuyama, S. und Mizushima, S., J. Bacteriol., 162(3), 1196–1202, 1985) wurde unter Ausnutzung der Temperaturempfindlichkeit des vorstehend genannten Plasmids pTSmalK-A124T durchgeführt.
-
Kurz gesagt wurde E. coli W3100 (tyrA) nach dem Verfahren von Hanahan et al. (J. Mol. Biol., 166, 557–580) unter Verwendung von pTSmalK-A124T transformiert. Eine Kolonie, die nach der Kultivierung bei 30°C auftrat, wurde in 5 ml eines L-Mediums, das 50 μg/ml Ampicillin enthielt, in einem Teströhrchen eingeimpft und bei 30°C über Nacht unter Schütteln kultiviert. Die Kulturbrühe wurde 103- bis 109-fach verdünnt, und 0,1 ml der Verdünnung wurde auf L-Agarmedium, das 50 μg/ml Ampicillin enthielt, aufgetragen und über Nacht bei 42°C kultiviert. Eine auftretende Kolonie wurde in 5 ml L-Medium in einem Teströhrchen eingeimpft und über Nacht bei 30°C unter Schütteln kultiviert. Diese Kulturbrühe in einem Volumen von 0,1 ml wurde in 5 ml L-Medium in einem Teströhrchen eingeimpft und bei 37–42°C während 3–4 Stunden unter Schütteln kultiviert. Diese Kulturbrühe wurde 103- bis 107-fach verdünnt, und 0,1 ml der Verdünnung wurde auf L-Agarmedium aufgetragen und über Nacht bei 37–42°C kultiviert. Die Ampicillinempfindlichkeit einer auftretenden Kolonie wurde bestätigt.
-
Der Mutationspunkt des mit dem Zielgen substituierten Stammes wurde wie folgt bestätigt. Eine PCR wurde unter Einsatz der vorstehend genannten Kolonie als Matrize und Pyrobest-DNA-Polymerase durchgeführt. Primer 1 und Primer 2 wurden als Primer eingesetzt, und die PCR wurde nach der dem Enzym beigefügten Anweisung durchgeführt. Nach dem vollständigen Ablauf der Reaktion wurde das Reaktionsgemisch einer Gelfiltration unterworfen, um die restlichen Primer aus dem Reaktionsgemisch zu entfernen. Die verwendete Säule war MicroSpinTM S-400HR (hergestellt von Amersham Pharmacia Biotech), und es wurde das Verfahren gemäß der der Säule beigefügten Anweisung durchgeführt. Das erhaltene PCR-Produkt war ein mutiertes malK-Gen des malK-Gen-substituierten Stammes. Die Nucleotidsequenz dieses Gens wurde hauptsächlich in dem Bereich bestimmt, welcher den Mutationspunkt enthielt. Es wurde bestätigt, dass der mutierte Stamm die gewünschte Mutation in dem malK-Gen enthielt. Der Stamm wurde E. coli W3100 (tyrA)malK1 genannt.
-
Das Wachstum von E. coli W3100 (tyrA)malK1 in einem Medium, das aus M9-Medium bestand, dem 0,05% Glucose und 0,45% Maltose beigefügt war, wurde durch OD-Messung überwacht (2). E. coli W3100 (tyrA) wurde als Kontrolle verwendet. Aus den in 2 gezeigten Ergebnissen geht hervor, dass obwohl das Zweiphasenwachstum, das heißt die so genannte Diauxie, für E. coli W3100 (tyrA) beobachtet wurde, ein solches Zweiphasenwachstum für den Stamm mit der eingeführten malK-Mutation, E. coli W3100 (tyrA)malK1, nicht beobachtet wurde. Es wurde also gefunden, dass aufgrund der Einführung der malK-Mutation der Induktorausschluss nicht verursacht wurde und Maltose gleichzeitig mit der Glucoseassimilation assimiliert wurde.
-
Beispiel 2: Einführen einer Mutation in das crr-Gen und Bestätigung der Verbesserung der Maltoseassimilationseigenschaft
-
In diesem Beispiel wurde entschieden, eine Mutation in das crr-Gen einzuführen, um die Wechselwirkung zwischen dem MalK-Protein und dem crr-Genprodukt IIAGlc zu verringern.
-
Eine Kolonie von E. coli W3100 wurde in 5 ml L-Medium eingeimpft und über Nacht unter Schütteln kultiviert. Aus den erhaltenen Zellen wurde chromosomale DNA unter Einsatz des Wizard Genomic DNA Purification Kit (Promega) hergestellt. Die PCR wurde unter Verwendung der vorstehend genannten chromosomalen DNA als Matrize und den folgenden Primern durchgeführt.
-
-
-
Die PCR wurde unter Einsatz der Pyrobest DNA Polymerase, hergestellt von Takara Shuzo, gemäß der dem Enzym beigefügten Anweisung durchgeführt. Nach dem vollständigen Ablauf der Reaktion wurde das Amplifikationsprodukt in dem Reaktionsgemisch abgestumpft und unter Verwendung des BKL-Kits von Takara Shuzo phosphoryliert. Das erhaltene amplifizierte Fragment wurde unter Verwendung des Ligation Kit ver.2 (Takara Shuzo) an pMW219 (Nippon Gene) ligiert, das mit einem Restriktionsenzym SmaI (Takara Shuzo) behandelt und dann dephosphoryliert wurde. E. coli JM109 wurde mit diesem Ligationsreaktionsgemisch gemäß dem Verfahren von Hanahan et al. transformiert. Die Selektion der Transformanten wurde auf einem L-Agarmedium durchgeführt, das 25 μg/ml Kanamycin (Km), 0,2 mM IPTG (Isopropyl-1-thio-β-D-galactopyranosid) und 40 μg/ml X-gal (5-Brom-4-chlor-3-indolyl-β-D-galactopyranosid) enthielt. Aus den Transformanten wurde ein Plasmid auf herkömmliche Weise extrahiert, und die Nucleotidsequenz des eingeführten Fragments wurde bestimmt, um zu bestätigen, dass das crr-Gen in die Sma-I-Stelle von pMW219 eingeführt worden war. Dieses Plasmid wurde pMWcrr genannt.
-
Die Nucleotidsubstitution des crr-Gens auf pMWcrr wurde wie folgt durchgeführt. Es wurde entschieden, die Nucleotidsubstitution von A gegen G in der Position 226 einzuführen (Substitution von Thr gegen den Ala-Rest in Position 76, Zeng, G. A. et al., Mutational analysis of the enzyme IIIGlc des Phosphoenolpyruvatphosphotransferasesystems in Escherichia coli, Res. Microbiol., 143, 252–261, 1992). Die Positionen in den DNA-Nucleotidsequenzen, die hier verwendet werden, sind von dem A des Initiationscodons ATG, welches als das erste Nucleotid genommen wurde, aus nummeriert, und die Positionen der Aminosäurereste sind von dem Met-Rest, welcher dem vorstehend genannten Initiationscodon entspricht und als der erste Aminosäurerest angenommen wurde, aus nummeriert.
-
Zuerst wurde die Substitution des Nucleotids auf dem Plasmid unter Verwendung von QuickChangeTM Site-Directed Mutagenesis Kit (STRATAGENE) durchgeführt. Die Sequenzen der für die Einführung der crr-Mutation eingesetzten Primer sind nachstehend gezeigt.
-
-
-
Die Einführung der Mutation wurde durch Bestimmen der Nucleotidsequenz der betreffenden Stelle gemäß der dem vorstehend genannten Kit beigefügten Anweisung bestätigt. Das hergestellte Plasmid wurde pMWcrr-A76T genannt. Das Plasmid wurde mit den Restriktionsenzymen EcoR I und Xba I (beide von Takara Shuzo) verdaut und zwischen dieselben Restriktionsenzymschnittstellen des Plasmidvektors pMAN997 (vergleiche die Internationale Patentveröffentlichung Nr.
WO 99/03988 ) mit einem temperaturempfindlichen Replikationsursprung ligiert.
-
Das hergestellte Plasmid wurde pMANcrr-A76T genannt. Die homologe Rekombination wurde für das crr-Gen auf dem Chromosom von E. coli W3100 (tyrA) unter Verwendung dieses Plasmids durch dasselbe Verfahren wie bei der Herstellung des malK-Mutantenstammes durchgeführt, und auf diese Weise wurde ein Stamm erhalten, in den eine Mutation in dem crr-Gen eingeführt worden war.
-
Die Mutationsstelle in dem Stamm mit dem substituierten Zielgen wurde auf dieselbe Weise wie bei dem malK-Mutantenstamm bestätigt. Die PCR wurde unter Einsatz einer Ampicillinresistenten Kolonie als Matrize und Ex Taq Polymerase (hergestellt von Takara Shuzo) durchgeführt. Primer 5 und Primer 6 wurden als die Primer verwendet, und die PCR wurde gemäß der dem Enzym beigefügten Anweisung durchgeführt. Nach dem vollständigen Ablauf der Reaktion wurde das Reaktionsgemisch einer Gelfiltration unterworfen, um die restlichen Primer in dem Reaktionsgemisch zu entfernen. Die verwendete Säule war MicroSpin TM S-400HR (hergestellt von Amersham Pharmacia Biotech), und das Verfahren wurde gemäß der der Säule beigefügten Anweisung durchgeführt. Das erhaltene PCR-Produkt war ein mutiertes crr-Gen des Stammes mit dem substituierten crr-Gen. Die Nucleotidsequenz dieses Gens wurde hauptsächlich für die Region, welche die Mutationsstelle enthielt, bestimmt. Der mutierte Stamm, für den bestätigt wurde, dass die vorstehend beschriebene gewünschte Mutation eingeführt worden war, wurde E. coli W3100 (tyrA)crr3 genannt.
-
Das Wachstum von E. coli W3100 (tyrA)crr3 in einem Medium, das aus M9-Medium, versetzt mit 0,05% Glucose und 0,45% Maltose, bestand, wurde durch OD-Messung überwacht (3). E. coli W3100 (tyrA) wurde als Kontrolle verwendet. 3 zeigt, dass obwohl ein Zweiphasenwachstum, das heißt die so genannte Diauxie, für E. coli W3100 (tyrA) beobachtet wurde, ein solches Zweiphasenwachstum für den Stamm E. coli W3100 (tyrA)crr3, in den die crr-Mutation eingeführt worden war, nicht beobachtet wurde. Das heißt, es wurde gefunden, dass aufgrund der Einführung der crr-Mutation der Induktorausschluss nicht verursacht wurde und Maltose gleichzeitig mit der Glucoseassimilation assimiliert wurde. Dasselbe wurde beobachtet, wenn die Nucleotidsubstitution von A gegen G in Position 139 (Substitution von Ser gegen Gly in Position 47) eingeführt wurde.
-
Beispiel 3: Herstellung eines Glucoseanalogon-resistenten Stammes, Identifikation der Mutationsstelle des resistenten Stammes und Einführen der Mutation in E. coli W3100 (tyrA)
-
Eine Kolonie von E. coli W3100 (tyrA) wurde in 5 ml eines L-Mediums in einem Teströhrchen eingeimpft und über Nacht unter Schütteln kultiviert. Die kultivierten Zellen wurden zweimal mit 5 ml physiologischer Kochsalzlösung gewaschen und in demselben Volumen einer physiologischen Kochsalzlösung suspendiert. Diese Suspension wurde in einer Menge von 0,1 ml auf M9-Agarmedium, welches Maltose als Kohlenstoffquelle enthielt, aufgetragen, und die Oberfläche wurde getrocknet. Eine Impföse mit 2-Deoxyglucose wurde auf die Platte gegeben, und die Zellen wurden. bei 37°C während zwei oder drei Tagen kultiviert. E. coli W3100 (tyrA) kann Maltose als Kohlenstoffquelle nutzen, wenn jedoch ein Glucoseanalogon, wie 2-Deoxyglucose, vorhanden ist, leidet dieser Stamm an der Repression und kann nicht mehr wachsen. In diesem Fall wird um den Punkt, auf den das Glucoseanalogon als Zentrum gegeben worden ist, ein Wachstumsinhibitionskreis gebildet. Wenn die Kultivierung während zwei oder drei Tagen durchgeführt wird, treten Kolonien, die wachsen können, in dem Inhibitionskreis mit einer bestimmten Häufigkeit auf. Glucoseanalogon-resistente Stämme wurden anhand dieses Phänomens erhalten.
-
Die Mutation des malK-Gens von malK#1 und malK#2 wurde unter den Glucoseanalogon-resistenten Stämmen untersucht. Die Mutationsstellen wurden wie folgt bestätigt. Eine Kolonie jedes Stammes wurde gebildet, und die PCR wurde unter Verwendung der Kolonie als Matrize und von Pyrobest DNA Polymerase durchgeführt. PCR wurde unter Verwendung von Primer 1 und Primer 2 als Primer gemäß der dem Enzym beigefügten Anweisung durchgeführt. Nach dem vollständigen Ablauf der Reaktion wurde das Reaktionsgemisch einer Gelfiltration unterworfen, um die restlichen Primer in dem Reaktionsgemisch zu entfernen. Die verwendete Säule war MicroSpinTM S-400HR (hergestellt von Amersham Pharmacia Biotech), und das Verfahren wurde gemäß der der Säule beigefügten Anweisung durchgeführt. Die erhaltenen PCR-Produkte waren malK-Gene des Glucose-resistenten Stammes malK#1 und malK#2, und die Nucleotidsequenzen dieser Gene wurden bestimmt. In beiden Stämmen wurde eine Substitution gegen T in Position 980 und entsprechend eine Substitution von Gln gegen Leu in Position 327 gefunden. Dies ist eine bislang unbekannte Mutation. Die Mutation wurde L327Q-Mutation bezeichnet.
-
Die L327Q-Mutation wurde in E. coli W3100 (tyrA) durch das vorstehend beschriebene Verfahren eingeführt. Der erhaltene Stamm mit eingeführter Mutation wurde E. coli W3100 (tyrA)malK327 genannt. Auf ähnliche Weise wurde das Wachstum von E. coli W3100 (tyrA)malK327 in einem Medium, das aus M9-Medium, versetzt mit 0,05% Glucose und 0,45% Maltose, bestand, durch OD-Messung überwacht (4). E. coli W3100 (tyrA) wurde als Kontrolle verwendet. 4 zeigt, dass obwohl ein Zweiphasenwachstum, das heißt die so genannte Diauxie, für E. coli W3100 (tyrA) beobachtet wurde, ein solches Zweiphasenwachstum für den Stamm E. coli W3100 (tyrA)malK327 mit eingeführter malK-Mutation nicht beobachtet wurde. Es wurde also gefunden, dass aufgrund der Einführung der neuen malK-Mutation der Induktorausschluss nicht bewirkt wurde und Maltose gleichzeitig mit der Glucoseassimiliation assimiliert wurde.
-
Beispiel 4: Untersuchung der L-Aminosäureproduktion von malK-Mutantenstämmen
-
pVIC40 (
WO 90/04636 ), pCABD2 (
WO 95/16042 ) und pMGAL1 (offen gelegte
Japanische Patentanmeldung (Kokai) Nr. 5-344881 ) wurden in E. coli W3100 (tyrA)malK327 eingeführt, und die Fähigkeit jedes Stammes, L-Lysin, L-Threonin und L-Phenylalanin zu produzieren, wurde untersucht.
-
Das Plasmid pVIC40 enthält das Threoninoperon und kann aus E. coli VKPM B-3996 (hinterlegt bei dem Antibiotika-Forschungsinstitut der UDSSR (VNIIA) unter der Nummer RIA1867), der das Plasmid trägt (
WO 90/04636 ), hergestellt werden.
-
Das Plasmid pCABD2 enthält DNA (dapA·24), die für Dihydrodipicolinatsynthase (DDPS), die von Escherichia coli abgeleitet ist und eine Mutation zur Ausschaltung der Rückkopplungshemmung durch L-Lysin trägt, eine DNA (lysC·80), die für von Escherichia coli abgeleitete Aspartokinase III mit einer Mutation zur Ausschaltung der Rückkopplungshemmung durch L-Lysin codiert, eine DNA (dapB), die für Dihydrodipicolinatreduktase aus Escherichia coli codiert, und eine DNA (ddh), die für eine Diaminopimelatdehydrogenase aus Brevibacterium lactofermentum (
WO 95/16042 ) codiert.
-
Das Plasmid pMGAL1 enthält ein Gen, das für 3-Deoxy-D-arabinohepturonat-7-phosphatsynthase aus einem Escherichia-Bakterium mit ausgeschalteter Rückkopplungshemmung codiert, und ein Gen, das für Chorismatmutaseprephenatdehydratase aus Escherichia coli mit ausgeschalteter Rückkopplungshemmung codiert (offen gelegte
Japanische Patentanmeldung Nr. 5-344881 ).
-
E. coli W3100 (tyrA)malK327 wurde mit jedem Plasmid gemäß den Verfahren von Hanahan et al. transformiert. Jede erhaltene Transformante wurde in 5 ml eines L-Mediums, das 50 μg/ml Streptomycin enthielt, eingeimpft und über Nacht bei 37°C unter Schütteln kultiviert. Dann wurde die Kulturbrühe in einer Menge von 50 μl auf L-Agarmedium, das 50 μg/ml Streptomycin enthielt, aufgetragen und über Nacht bei 37°C kultiviert. Ein Aminosäureproduktionsmedium, das ein Gemisch von Glucose und Maltose (36 g/l Glucose, 5,8 g/l Maltose) als Kohlenstoffquelle enthielt, wurde in einem Volumen von 20 ml in einen 500 ml-Sakaguchikolben gegeben, und 1/8 der auf dem vorstehend genannten Agarmedium gewachsenen Zellen wurde abgeschabt und in das Medium eingeimpft. Nach dem vollständigen Ablauf der Kultivierung wurde die Konzentration jeder Aminosäure und der verbleibenden Glucose und Maltose bestimmt. Als Kontrollen wurden Transformanten eingesetzt, die durch Einführen jedes der Plasmide in E. coli W3100 (tyrA) erhalten wurden. Die Ergebnisse sind in Tabelle 1 gezeigt. Tabelle 1
| Stamm (Wirt/Plasmid) | Lys (g/l) | Thr (g/l) | Phe (g/l) | Verbliebene Glucose (g/l) | Verbliebene Maltose (g/l) |
| W3110(tyrA)/pRS | 0,00 | 0,00 | 0,91 | 0,0 | 5,8 |
| W3110(tyrA)malK327/pRS | 0,00 | 0,00 | 0,86 | 0,0 | 0,0 |
| W3110(tyrA)/pCABD2 | 9,55 | - | - | 0,0 | 5,6 |
| W3110(tyrA)malK327/pCABD2 | 10,80 | - | - | 0,0 | 0,0 |
| W3110(tyrA)/pVIC40 | - | 8,48 | - | 0,0 | 4,1 |
| W3110(tyrA)malK327/pVIC40 | - | 8,62 | - | 0,0 | 0,0 |
| W3110(tyrA)/pMGAL1 | - | - | 4,57 | 0,0 | 5,7 |
| W3110(tyrA) malK327/pMGALl | - | - | 4,69 | 1,2 | 0,0 |
-
-: Nicht bestimmt, pRS: Vektor (die anfängliche Konzentration von Glucose und Maltose war 36 g/l beziehungsweise 5,8 g/l, und die Kultivierungsdauer war 14 Stunden)
Lys: Lysin; Thr: Threonin; Phe: Phenylalanin
-
Wenn E. coli W3100 (tyrA) als Wirt eingesetzt wurde, wurde Maltose nicht assimiliert, Glucose wurde jedoch verbraucht.
-
Andererseits wurde, wenn E. coli W3100 (tyrA)malK327 als Wirt eingesetzt wurde, Maltose innerhalb einer ähnlichen Kultivierungsdauer assimiliert, sodass gefunden wurde, dass der Verbrauch von Maltose von der Glucoserepression nicht betroffen war.
-
Außerdem zeigten die Stämme von E. coli W3100 (tyrA)malK327, welche jeweils pVIC40, pCABD2 und pMGAL1 enthielten, im Vergleich zu den Stämmen von E. coli W3100(tyrA), welche die jeweiligen Plasmide enthielten, verbesserte Fähigkeit zur Produktion von L-Lysin, L-Threonin und L-Phenylalanin.
-
-