DECLARAÇÃO QUANTO A LISTAGEM DA SEQUÊNCIA.
[001]A Listagem da sequência associada com este pedido é fornecida no formato de texto em vez de uma cópia de papel, e é desse modo incorporado pela referência no Relatório Descritivo. O nome do arquivo de texto contendo a Listagem da sequência é 480239_403PC_SEQUENCE_LISTING.txt. O arquivo de texto tem 515 kb, foi criado no dia 4 de abril de 2007, e está sendo entregue eletronicamente via a EFS-web, simultaneamente com o arquivamento do Relatório Descritivo.
ANTECEDENTES.
Campo da técnica.
[002]Presente invenção se refere geralmente a composições compreenden-do as combinações antigênicas e/ou imunogênicas dos antígenos de Mycobacterium tuberculosis e his uso no diagnóstico, tratamento, e prevenção da tuberculose.
Descrição da Técnica Relacionada.
[003]A tuberculose é uma doença contagiosa crônica causada pela infecção com Mycobacterium tuberculosis e outras espécies de Mycobacterium. É uma doença importante em países em desenvolvimento, bem como um problema crescente em áreas desenvolvidas do mundo, com vários milhões de novos casos cada ano.
[004]Embora a infecção possa ser assintomática por um período de tempo comsiderável, a doença é bem mais comumente manifestada como uma inflamação aguda dos pulmões, resultando em febre e uma tosse improdutiva. Se não tratada, resulta tipicamente em complicações sérias e morte.
[005]Embora a tuberculose possa ser geralmente controlada usando extensa terapia com antibiótico, tal tratamento não é suficiente para prevenir a disseminação da doença. Os indivíduos infectados podem ser assintomáticos, mas contagiosos, por algum tempo. Além disso, embora a obediência ao regime de tratamento seja crítica, o comportamento do paciente é difícil de controlar. Alguns pacientes não completam o curso do tratamento, o que pode levar ao tratamento ineficaz e o desenvolvimento de resistência à droga.
[006]Para controlar a disseminação da tuberculose, a vacinação eficaz e precisa e o diagnóstico precoce da doença são críticos. Atualmente, a vacinação com bactérias vivas é o bem mais amplamente método usado para induzir a imunidade protetora. O Mycobacterium bem mais comum empregado com esta finalidade é Bacillus Calmette-Guerin (BCG), uma cepa avirulenta de Mycobacterium bovis.
[007]Entretanto, a segurança e a eficácia da BCG são uma fonte de contro-vérsia e alguns países, tais como os Estados Unidos, não vacinam o grande público com este agente.
[008]O diagnóstico da tuberculose é comumente atingido usando um teste de pele, que envolve a exposição intradérmica à tuberculina PPD (derivado de prote-ína purificada). As respostas de célula T específicas para o antígeno resultam no endurecimento mensurável no sítio de injeção por 48-72 horas depois da injeção, que indica a exposição a antígenos de micobactéria. A sensibilidade e a especifici-dade foram, entretanto, problemáticas, e os indivíduos vacinados com BCG não po-dem ser distinguidos dos indivíduos infectados.
[009]Consequentemente existe uma necessidade de reagentes melhorados e métodos de diagnosticar, prevenir e tratar a tuberculose. A presente invenção cumpre estas necessidades e oferece outras vantagens relacionadas.
BREVE SUMÁRIO.
[0010]Presente invenção se refere geralmente a composições compreen-dendo pelo menos dois antígenos heterólogos, polipeptídeos de fusão compreen-dendo os antígenos e polinucleotídeos codificando os antígenos, onde os antígenos são da espécie Mycobacterium, particularmente Mycobacterium tuberculosis. A pre-sente invenção também se refere métodos para utilização dos polipeptídeos e os polinucleotídeos de acordo com a invenção no diagnóstico, tratamento e prevenção da infecção por Mycobacterium. Os antígenos de acordo com a invenção, quando empregados em combinação e/ou como polipeptídeos de fusão ou polinucleotídeos tal como aqui descrito, oferecem níveis melhorados e inesperados de imunogenici- dade, resultando em redução na carga bacteriana no pulmão, e assim serem particu-larmente úteis no contexto do desenvolvimento da vacina.
[0011]Por exemplo, em um aspecto de acordo com a invenção, são forneci-das composições compreendendo um imunoestimulante e uma combinação de dois ou mais antígenos de Mycobacterium tuberculosis, ou fragmentos imunogênicos do mesmo, em que os antígenos são selecionados do grupo consistindo de Rv0164 (SEQ ID NO: 1), Rv0496 (SEQ ID NO: 6), Rv2608 (SEQ ID NO: 26), Rv3020 (SEQ ID NO: 36), Rv3478 (SEQ ID NO: 41), Rv3619 (SEQ ID NO: 46), Rv3620 (SEQ ID NO: 51), RV1738 (SEQ ID NO: 11), Rv1813 (SEQ ID NO: 16), Rv3810 (SEQ ID NO: 56), Rv2389 (SEQ ID NO: 21), Rv2866 (SEQ ID NO: 31), Rv3876 (SEQ ID NO: 61), Rv0054 (SEQ ID NO: 100), Rv0410 (SEQ ID NO: 106), Rv0655 (SEQ ID NO: 112), Rv0831 (SEQ ID NO: 115), Rv1009 (SEQ ID NO: 118), Rv1099 (SEQ ID NO: 121), Rv1240 (SEQ ID NO: 124), Rv1288 (SEQ ID NO: 127), Rv1410 (SEQ ID NO: 130), Rv1569 (SEQ ID NO: 133), Rv1789 (SEQ ID NO: 136), Rv1818 (SEQ ID NO: 139), Rv1860 (SEQ ID NO: 142), Rv1886 (SEQ ID NO: 145), Rv1908 (SEQ ID NO: 148), Rv2220 (SEQ ID NO: 154), Rv2032 (SEQ ID NO: 151), Rv2623 (SEQ ID NO: 160), Rv2875 (SEQ ID NO: 163), Rv3044 (SEQ ID NO: 166), Rv3310 (SEQ ID NO: 169), Rv3881 (SEQ ID NO: 178), Rv0577 (SEQ ID NO: 184), Rv1626 (SEQ ID NO: 187), Rv0733 (SEQ ID NO: 190), Rv2520 (SEQ ID NO: 193), Rv1253 (SEQ ID NO: 196), Rv1980 (SEQ ID NO: 199), Rv3628 (SEQ ID NO: 202) Rv1884 (SEQ ID NO: 205), Rv3872 (SEQ ID NO: 208), Rv3873 (SEQ ID NO: 211), Rv1511 (SEQ ID NO: 214) e Rv3875 (SEQ ID NO: 292) e antígenos possuindo pelo menos 80 %, 90 % ou 95 % de identidade com quaisquer das sequências precedentes.
[0012]Em certas modalidades, a combinação de dois ou mais antígenos é selecionada do grupo consistindo de: (a) uma combinação compreendendo Rv1813 (SEQ ID NO: 16); Rv3620 (SEQ ID NO: 51) e Rv2608 (SEQ ID NO: 26); (b) uma combinação compreendendo Rv2608 (SEQ ID NO: 26) e Rv3619 (SEQ ID NO: 46); e (c) uma combinação compreendendo Rv3478 (SEQ ID NO: 41) e Rv3619 (SEQ ID NO: 46).
[0013]Em uma modalidade particular, a composição de (a) acima, compre-endendo Rv2608 (SEQ ID NO: 26), Rv1813 (SEQ ID NO: 16) e Rv3620 (SEQ ID NO: 51), também compreende um ou mais antígenos selecionados do grupo consistindo de: Rv1886 (SEQ ID NO: 145), Rv2389 (SEQ ID NO: 21), Rv3478 (SEQ ID NO: 41), Rv2875 (SEQ ID NO: 163), Rv2220 (SEQ ID NO: 154), Rv0733 (SEQ ID NO: 190), Rv0577 (SEQ ID NO: 184), Rv3044 (SEQ ID NO: 166), Rv1626 (SEQ ID NO: 187), Rv3619 (SEQ ID NO: 46) e Rv3020 (SEQ ID NO: 36).
[0014]Em uma modalidade mais particular, a composição compreende Rv1813 (SEQ ID NO: 16); Rv3620 (SEQ ID NO: 51), Rv2608 (SEQ ID NO: 26) e Rv2389 (SEQ ID NO: 21).
[0015]Em uma modalidade particular relacionada, a composição compreende Rv2608 (SEQ ID NO: 26); Rv1813 (SEQ ID NO: 16), Rv3620 (SEQ ID NO: 51) e Rv3619 (SEQ ID NO: 46).
[0016]Em certas outras modalidades de acordo com a invenção, a composi-ção de (b) acima, compreendendo Rv2608 (SEQ ID NO: 26) e Rv3619 (SEQ ID NO: 46), também compreende um ou mais antígenos selecionados do grupo consistindo de: Rv1886 (SEQ ID NO: 145), Rv2389 (SEQ ID NO: 21), Rv1813 (SEQ ID NO: 16), Rv2875 (SEQ ID NO: 163), Rv2220 (SEQ ID NO: 154), Rv0733 (SEQ ID NO: 190), Rv0577 (SEQ ID NO: 184), Rv3044 (SEQ ID NO: 166), Rv1626 (SEQ ID NO: 187), Rv3620 (SEQ ID NO: 51), Rv3478 (SEQ ID NO: 41), e Rv3020 (SEQ ID NO: 36).
[0017]Em uma modalidade particular, a composição compreende Rv2608 (SEQ ID NO: 26), Rv3619 (SEQ ID NO: 46), e Rv1886 (SEQ ID NO: 145).
[0018]Em outra modalidade particular, a composição também compreende um ou mais antígenos selecionados do grupo consistindo de: Rv2389 (SEQ ID NO: 21), Rv1813 (SEQ ID NO: 16), Rv2875 (SEQ ID NO: 163), Rv2220 (SEQ ID NO: 154), Rv0733 (SEQ ID NO: 190), Rv0577 (SEQ ID NO: 184), Rv3044 (SEQ ID NO: 166), Rv1626 (SEQ ID NO: 187), Rv3620 (SEQ ID NO: 51) e Rv3020 (SEQ ID NO: 36).
[0019]Em uma modalidade mais particular, a composição compreende Rv2608 (SEQ ID NO: 26), Rv3619 (SEQ ID NO: 46), Rv1813 (SEQ ID NO: 16) e Rv3620 (SEQ ID NO: 51).
[0020]Em certas outras modalidades de acordo com a invenção, a composi- çãode (c) acima, compreendendo Rv3478 (SEQ ID NO: 41) e Rv3619 (SEQ ID NO: 46), também compreende um ou mais antígenos selecionados do grupo consistindo de: Rv1886 (SEQ ID NO: 145), Rv2389 (SEQ ID NO: 21), Rv1813 (SEQ ID NO: 16), Rv2875 (SEQ ID NO: 163), Rv2220 (SEQ ID NO: 154), Rv0733 (SEQ ID NO: 190), Rv0577 (SEQ ID NO: 184), Rv3044 (SEQ ID NO: 166), Rv1626 (SEQ ID NO: 187), Rv3620 (SEQ ID NO: 51), Rv2608 (SEQ ID NO: 26), e Rv3020 (SEQ ID NO: 36).
[0021]Em uma modalidade particular, a composição compreende Rv3478 (SEQ ID NO: 41), Rv3619 (SEQ ID NO: 46) e Rv1886 (SEQ ID NO: 145).
[0022]Em outra modalidade, a combinação também compreende um ou mais antígenos selecionados do grupo consistindo de: Rv2389 (SEQ ID NO: 21), Rv1813 (SEQ ID NO: 16), Rv2875 (SEQ ID NO: 163), Rv2220 (SEQ ID NO: 154), Rv0733 (SEQ ID NO: 190), Rv0577 (SEQ ID NO: 184), Rv3044 (SEQ ID NO: 166), Rv1626 (SEQ ID NO: 187) e Rv3020 (SEQ ID NO: 36).
[0023]A combinação de dois ou mais antígenos aqui descritos pode incluir uma combinação de dois ou mais antígenos recombinantes separados, ou fragmen-tos antigênicos/imunogênicos dos mesmos. Altemativamente, os dois ou mais antí-genos, ou fragmentos antigênicos/imunogênicos do mesmo, podem ser covalente- mente ligados na forma de um polipeptídeo de fusão.
[0024]De acordo com outro aspecto de acordo com a invenção, existem po-lipeptídeos de fusão isolados fornecidos compreendendo uma combinação de dois ou mais antígenos de Mycobacterium tuberculosis covalentemente ligados, ou frag-mentos imunogênicos do mesmo, em que os antígenos são selecionados do grupo consistindo de Rv0164 (SEQ ID NO: 1), Rv0496 (SEQ ID NO: 6), Rv2608 (SEQ ID NO: 26), Rv3020 (SEQ ID NO: 36), Rv3478 (SEQ ID NO: 41), Rv3619 (SEQ ID NO: 46), Rv3620 (SEQ ID NO: 51), RV1738 (SEQ ID NO: 11), Rv1813 (SEQ ID NO: 16), Rv3810 (SEQ ID NO: 56), Rv2389 (SEQ ID NO: 21), Rv2866 (SEQ ID NO: 31), Rv3876 (SEQ ID NO: 61), Rv0054 (SEQ ID NO: 100), Rv0410 (SEQ ID NO: 106), Rv0655 (SEQ ID NO: 112), Rv0831 (SEQ ID NO: 115), Rv1009 (SEQ ID NO: 118), Rv1099 (SEQ ID NO: 121), Rv1240 (SEQ ID NO: 124), Rv1288 (SEQ ID NO: 127), Rv1410 (SEQ ID NO: 130), Rv1569 (SEQ ID NO: 133), Rv1789 (SEQ ID NO: 136), Rv1818 (SEQ ID NO: 139), Rv1860 (SEQ ID NO: 142), Rv1886 (SEQ ID NO: 145), Rv1908 (SEQ ID NO: 148), Rv2220 (SEQ ID NO: 154), Rv2032 (SEQ ID NO: 151), Rv2623 (SEQ ID NO: 160), Rv2875 (SEQ ID NO: 163), Rv3044 (SEQ ID NO: 166), Rv3310 (SEQ ID NO: 169), Rv3881 (SEQ ID NO: 178), Rv0577 (SEQ ID NO: 184), Rv1626 (SEQ ID NO: 187), Rv0733 (SEQ ID NO: 190), Rv2520 (SEQ ID NO: 193), Rv1253 (SEQ ID NO: 196), Rv1980 (SEQ ID NO: 199), Rv3628 (SEQ ID NO: 202) Rv1884 (SEQ ID NO: 205), Rv3872 (SEQ ID NO: 208), Rv3873 (SEQ ID NO: 211), Rv1511 (SEQ ID NO: 214), e Rv3875 (SEQ ID NO: 292) e antígenos possuindo pelo menos 80 %, 90 % ou 95 % de identidade com quaisquer das sequências preceden- tes.
[0025]Em certas modalidades, o polipeptídeo de fusão compreende uma combinação de antígenos covalentemente ligados selecionado do grupo consistindo de: (d) uma combinação compreendendo Rv1813 (SEQ ID NO: 16); Rv3620 (SEQ ID NO: 51) e Rv2608 (SEQ ID NO: 26); (e) uma combinação compreendendo Rv2608 (SEQ ID NO: 26) e Rv3619 (SEQ ID NO: 46); e (f) uma combinação compreendendo Rv3478 (SEQ ID NO: 41) e Rv3619 (SEQ ID NO: 46).
[0026]Em uma modalidade particular, o polipeptídeo de fusão de (a) acima, compreendendo Rv2608 (SEQ ID NO: 26), Rv1813 (SEQ ID NO: 16) e Rv3620 (SEQ ID NO: 51), também compreende um ou mais antígenos selecionados do grupo con-sistindo de: Rv1886 (SEQ ID NO: 145), Rv2389 (SEQ ID NO: 21), Rv1813 (SEQ ID NO: 16), Rv2875 (SEQ ID NO: 163), Rv2220 (SEQ ID NO: 154), Rv0733 (SEQ ID NO: 190), Rv0577 (SEQ ID NO: 184), Rv3044 (SEQ ID NO: 166), Rv1626 (SEQ ID NO: 187), Rv3619 (SEQ ID NO: 46), Rv3478 (SEQ ID NO: 41) e Rv3020 (SEQ ID NO: 36).
[0027]Em uma modalidade mais particular, o polipeptídeo de fusão compre-ende Rv1813 (SEQ ID NO: 16); Rv3620 (SEQ ID NO: 51); Rv2608 (SEQ ID NO: 26) e Rv2389 (SEQ ID NO: 21).
[0028]Em uma modalidade particular relacionada, o polipeptídeo de fusão compreende Rv1813 (SEQ ID NO: 16); Rv3620 (SEQ ID NO: 51); Rv2608 (SEQ ID NO: 26) e Rv3619 (SEQ ID NO: 46).
[0029]Em certas outras modalidades de acordo com a invenção, o polipeptí-deo de fusão de (b) acima, compreendendo Rv2608 (SEQ ID NO: 26) e Rv3619 (SEQ ID NO: 46), também compreende um ou mais antígenos selecionados do grupo consistindo de: Rv1886 (SEQ ID NO: 145), Rv2389 (SEQ ID NO: 21), Rv1813 (SEQ ID NO: 16), Rv2875 (SEQ ID NO: 163), Rv2220 (SEQ ID NO: 154), Rv0733 (SEQ ID NO: 190), Rv0577 (SEQ ID NO: 184), Rv3044 (SEQ ID NO: 166), Rv1626 (SEQ ID NO: 187), Rv3620 (SEQ ID NO: 51), Rv3478 (SEQ ID NO: 41), e Rv3020 (SEQ ID NO: 36).
[0030]Em uma modalidade particular, o polipeptídeo de fusão compreende Rv2608 (SEQ ID NO: 26), Rv1813 (SEQ ID NO: 16), Rv3619 (SEQ ID NO: 46), e Rv1886 (SEQ ID NO: 145).
[0031]Em outra modalidade particular, o polipeptídeo de fusão também com-preende um ou mais antígenos selecionados do grupo consistindo de: Rv2389 (SEQ ID NO: 21), Rv1813 (SEQ ID NO: 16), Rv2875 (SEQ ID NO: 163), Rv2220 (SEQ ID NO: 154), Rv0733 (SEQ ID NO: 190), Rv0577 (SEQ ID NO: 184), Rv3044 (SEQ ID NO: 166), Rv1626 (SEQ ID NO: 187), Rv3620 (SEQ ID NO: 51) e Rv3020 (SEQ ID NO: 36).
[0032]Em uma modalidade mais particular, o polipeptídeo de fusão compre-ende Rv2608 (SEQ ID NO: 26), Rv3619 (SEQ ID NO: 46), Rv1813 (SEQ ID NO: 16) e Rv3620 (SEQ ID NO: 51).
[0033]Em certas outras modalidades de acordo com a invenção, o polipeptí-deo de fusãode (c) acima, compreendendo Rv3478 (SEQ ID NO: 41) e Rv3619 (SEQ ID NO: 46), também compreende um ou mais antígenos selecionados do grupo consistindo de: Rv1886 (SEQ ID NO: 145), Rv2389 (SEQ ID NO: 21), Rv1813 (SEQ ID NO: 16), Rv2875 (SEQ ID NO: 163), Rv2220 (SEQ ID NO: 154), Rv0733 (SEQ ID NO: 190), Rv0577 (SEQ ID NO: 184), Rv3044 (SEQ ID NO: 166), Rv1626 (SEQ ID NO: 187), Rv3620 (SEQ ID NO: 51), Rv2608 (SEQ ID NO: 26), e Rv3020 (SEQ ID NO: 36).
[0034]Em uma modalidade particular, o polipeptídeo de fusão compreende Rv3478 (SEQ ID NO: 41), Rv3619 (SEQ ID NO: 46) e Rv1886 (SEQ ID NO: 145).
[0035]Em outra modalidade, o polipeptídeo de fusão também compreende um ou mais antígenos selecionados do grupo consistindo de: Rv2389 (SEQ ID NO: 21), Rv1813 (SEQ ID NO: 16), Rv2875 (SEQ ID NO: 163), Rv2220 (SEQ ID NO: 154), Rv0733 (SEQ ID NO: 190), Rv0577 (SEQ ID NO: 184), Rv3044 (SEQ ID NO: 166), Rv1626 (SEQ ID NO: 187) e Rv3020 (SEQ ID NO: 36).
[0036]Em certas modalidades particulares, os polipeptídeos de fusão são fornecidos compreendendo uma sequência de aminoácidos selecionado do grupo consistindo de: ID83 (SEQ ID NO: 91), ID94 (SEQ ID NO: 95), ID93 (SEQ ID NO: 226), ID91 (SEQ ID NO: 236), ID71 (SEQ ID NO: 245), ID114 (SEQ ID NO: 251), ID125 (SEQ ID NO: 257).
[0037]De acordo com outro aspecto de acordo com a invenção, são forneci-dos polinucleotídeos isolados codificando quaisquer dos antígenos e/ou polipeptí-deos de fusão aqui descritos.
[0038]Será compreendido que, em muitas modalidades, as composições, os polipeptídeos e os polinucleotídeos de acordo com a invenção são preferivelmente formulados em combinação com um ou mais imunoestimulantes para melhorar a resposta imune produzida pelos antígenos aqui descritos.
[0039]Numerosos imunoestimulantes e sistemas adjuvantes são conhecidos e disponíveis na técnica e podem ser usados no contexto da presente invenção, os exemplos ilustrativos dos quais incluem AS-2, ENHANZYN®, MPL®, 3D-MPL®, IFA, QS21, CWS, TDM, AGPs, Oligonucleotídios contendo CpG, agonistas dos Toll-like receptor (por exemplo, agonistas de TLR9, agonistas de TLR7, agonistas de TLR7/8, agonistas de TLR5, agonistas de TLR4, agonistas de TLR2, agonistas de TLR3, etc.), LeIF, saponinas, miméticos da saponina, e lipídio A biológico e sintético, imi- quimod, gardiquimod, resiquimod, polil:C, flagelina, ou uma combinação das mesmas.
[0040]Qs polinucleotídeos de fusão, polipeptídeos de fusão, ou composições de acordo com a invenção foram encontrados como sendo altamente antigênicos. Desse modo, de acordo com outro aspecto de acordo com a invenção, existem vacinas fornecidas e métodos relacionados para estimular uma resposta imune protetora em um paciente administrando uma quantidade eficaz de uma composição tal como as aqui descritas. Os polinucleotídeos isolados ou purificados podem ser usados para produzir antígenos de polipeptídeo de fusão recombinantes in vitro, que são então administrados como uma vacina. Alternativamente, os polinucleotídeos podem ser administrados diretamente a um paciente como uma vacina à base de DNA para causar a expressão do antígeno no paciente, e a indução subsequente de uma resposta imune anti-Mycobacterium tuberculosis.
[0041]Além disso, as composições, os polipeptídeos de fusão e os polinu-cleotídeos são úteis como ferramentas para diagnóstico em pacientes que podem ter sido infectados com Mycobacterium. Por exemplo, as composições, os polipeptídeos de fusão, e os polinucleotídeos de acordo com a invenção podem ser usados em testes in vitro e in vivo para detectar anticorpos humorais ou imunidade mediada por célula contra Mycobacterium tuberculosis para o diagnóstico da infecção, monitora-mento da progressão de doença e/ou avaliação de testes para cura.
[0042]Em uma modalidade, são fornecidos kits de diagnóstico para detectar a infecção de Mycobacterium tuberculosis em uma amostra biológica, compreendendo (a) um polipeptídeo compreendendo pelo menos uma porção imunogênica de um antígeno ou polipeptídeo de fusão aqui descrito, (b) um reagente de detecção.
[0043]Em outra modalidade, são fornecidos os métodos para detecção da presença da infecção por Mycobacterium tuberculosis em uma amostra biológica, compreendendo (a) contactar uma amostra biológica com um anticorpo monoclonal que se liga a um antígeno ou polipeptídeo de fusão aqui descrito; e (b) detectar na amostra biológica a presença de proteínas de Mycobacterium tuberculosis que ligam ao anticorpo monoclonal.
[0044]Em ainda outra modalidade, são fornecidos os métodos para detecção da infecção por Mycobacterium tuberculosis em uma amostra biológica, compreen-dendo (a) contactar a amostra biológica com uma combinação de antígeno ou poli-peptídeo de fusão tal como aqui descrito e (b) detectar na amostra biológica a pre-sença de anticorpos e/ou de células T que se ligam ao mesmo.
[0045]Em uma modalidade particular, são fornecidos os métodos para de-tecção da infecção por Mycobacterium tuberculosis em uma amostra biológica, com-preendendo (a) contactar a amostra biológica com uma combinação de dois ou mais antígenos selecionados do grupo consistindo de Rv0164 (SEQ ID NO: 1), Rv0496 (SEQ ID NO: 6), Rv2608 (SEQ ID NO: 26), Rv3020 (SEQ ID NO: 36), Rv3478 (SEQ ID NO: 41), Rv3619 (SEQ ID NO: 46), Rv3620 (SEQ ID NO: 51), RV1738 (SEQ ID NO: 11), Rv1813 (SEQ ID NO: 16), Rv3810 (SEQ ID NO: 56), Rv2389 (SEQ ID NO: 21), Rv2866 (SEQ ID NO: 31), Rv3876 (SEQ ID NO: 61), Rv0054 (SEQ ID NO: 100), Rv0410 (SEQ ID NO: 106), Rv0655 (SEQ ID NO: 112), Rv0831 (SEQ ID NO: 115), Rv1009 (SEQ ID NO: 118), Rv1099 (SEQ ID NO: 121), Rv1240 (SEQ ID NO: 124), Rv1288 (SEQ ID NO: 127), Rv1410 (SEQ ID NO: 130), Rv1569 (SEQ ID NO: 133), Rv1789 (SEQ ID NO: 136), Rv1818 (SEQ ID NO: 139), Rv1860 (SEQ ID NO: 142), Rv1886 (SEQ ID NO: 145), Rv1908 (SEQ ID NO: 148), Rv2220 (SEQ ID NO: 154), Rv2032 (SEQ ID NO: 151), Rv2623 (SEQ ID NO: 160), Rv2875 (SEQ ID NO: 163), Rv3044 (SEQ ID NO: 166), Rv3310 (SEQ ID NO: 169), e Rv3881 (SEQ ID NO: 178), Rv0577 (SEQ ID NO: 184), Rv1626 (SEQ ID NO: 187), Rv0733 (SEQ ID NO: 190), Rv2520 (SEQ ID NO: 193), Rv1253 (SEQ ID NO: 196), Rv1980 (SEQ ID NO: 199), Rv3628 (SEQ ID NO: 202) Rv1884 (SEQ ID NO: 205), Rv3872 (SEQ ID NO: 208), Rv3873 (SEQ ID NO: 211), Rv1511 (SEQ ID NO: 214) e Rv3875 (SEQ ID NO: 292), ou porções imunogênicas do mesmo; e (b) detectar na amostra biológica a presença de anticorpos e/ou de células T que se ligam ao mesmo.
[0046]Em uma modalidade particular, um método para detectar a infecção de Mycobacterium tuberculosis em uma amostra biológica compreende: contactar a amostra biológica com um polipeptídeo de fusão selecionado do grupo consistindo de: DID85 (SEQ ID NO: 265); DID92 (SEQ ID NO: 273); DID108 (SEQ ID NO: 283) e DID93 (SEQ ID NO: 291); e detectar na amostra biológica a presença de anticorpos e/ou de células T que se ligam ao mesmo.
[0047]Em outra modalidade particular, a invenção fornece kits para diagnós-tico para detectar a infecção de Mycobacterium tuberculosis em uma amostra bioló-gica, compreendendo: (a) uma combinação de dois ou mais antígenos selecionados do grupo consistindo de Rv0164 (SEQ ID NO: 1), Rv0496 (SEQ ID NO: 6), Rv2608 (SEQ ID NO: 26), Rv3020 (SEQ ID NO: 36), Rv3478 (SEQ ID NO: 41), Rv3619 (SEQ ID NO: 46), Rv3620 (SEQ ID NO: 51), RV1738 (SEQ ID NO: 11), Rv1813 (SEQ ID NO: 16), Rv3810 (SEQ ID NO: 56), Rv2389 (SEQ ID NO: 21), Rv2866 (SEQ ID NO: 31), Rv3876 (SEQ ID NO: 61), Rv0054 (SEQ ID NO: 100), Rv0410 (SEQ ID NO: 106), Rv0655 (SEQ ID NO: 112), Rv0831 (SEQ ID NO: 115), Rv1009 (SEQ ID NO: 118), Rv1099 (SEQ ID NO: 121), Rv1240 (SEQ ID NO: 124), Rv1288 (SEQ ID NO: 127), Rv1410 (SEQ ID NO: 130), Rv1569 (SEQ ID NO: 133), Rv1789 (SEQ ID NO: 136), Rv1818 (SEQ ID NO: 139), Rv1860 (SEQ ID NO: 142), Rv1886 (SEQ ID NO: 145), Rv1908 (SEQ ID NO: 148), Rv2220 (SEQ ID NO: 154), Rv2032 (SEQ ID NO: 151), Rv2623 (SEQ ID NO: 160), Rv2875 (SEQ ID NO: 163), Rv3044 (SEQ ID NO: 166), Rv3310 (SEQ ID NO: 169), e Rv3881 (SEQ ID NO: 178), Rv0577 (SEQ ID NO: 184), Rv1626 (SEQ ID NO: 187), Rv0733 (SEQ ID NO: 190), Rv2520 (SEQ ID NO: 193), Rv1253 (SEQ ID NO: 196), Rv1980 (SEQ ID NO: 199), Rv3628 (SEQ ID NO: 202) Rv1884 (SEQ ID NO: 205), Rv3872 (SEQ ID NO: 208), Rv3873 (SEQ ID NO: 211), Rv1511 (SEQ ID NO: 214) e Rv3875 (SEQ ID NO: 292), ou porções imunogê- nicas do mesmo; e (b) um reagente de detecção.
[0048]Em uma modalidade particular, um kit da presente invenção para de-tectar a infecção de Mycobacterium tuberculosis em uma amostra biológica compre-ende: um polipeptídeo de fusão selecionado do grupo consistindo de: DID85 (SEQ ID NO: 265), DID92 (SEQ ID NO: 273), DID108 (SEQ ID NO: 283) e DID93 (SEQ ID NO: 291), e um reagente de detecção.
BREVE DESCRIÇÃO DE VÁRIAS VISTAS DOS DESENHOS.
[0049]A figura 1 mostra que os níveis de IFN-y liberado pelo PBMC humano estimulado por atígeno. PBMC PPD’ e PPD+ são incubados por 72 h em um meio composto de, 10 pg/mL PHA, 10 pg/mL do lisado de Mtb, 50 pg/mL das proteínas recombinantes Mtb.
[0050]Média (MédiaAg - MédiaMeio) ± Média do Erro Padrão, são mostrados para PBMC PPD+ (n = 18) e PPD’ (n = 7).
[0051]A figura 2 mostra os níveis de esplenócitos TNF+ durante a estimula-ção in vitro por antígeno com diferentes proteínas recombinantes de Mtb. Os esple-nócitos de camundongos infectados com uma dose baixa de M. tuberculosis virulen-to H37Rv são coletados 4 semanas e 12 semanas depois da infecção e foram testa-dos para respostas da citoquina TNF específicas para o antígeno por ELISPOT. Os esplenócitos são incubados por 48 h em um meio composto de, 10 pg/mL do lisado de Mtb, ou 10 pg/mL das proteínas recombinantes Mtb. Os dados mostrados são a média ± SD (desvio padrão) (n = 2) em um experimento representativo. As figuras 3A-3D mostram a proteção contra infecção de M. tuberculosis e as respostas imunes específicas para o antígeno.
[0052]A figura 3A mostra o Logw CFU no pulmão de camundongos imuniza-dos depois de um teste de provocação com aerossol com M. tuberculosis. Os pulmões de camundongos (n = 7) imunizados com CpG, 3 diferentes antígenos Mtb Rv, ou uma combinação dos mesmos são coletados 4 semanas depois de um teste de provocação com aerossol com 50- 100 bacilos Mtb. As CFU foram contadas depois de 2 semanas de crescimento in vitro na placa de ágar-ágar. Os dados mostrados são a média ± média do erro padrão, de um experimento representativo. A figura 3B mostra as titulações de ponto final de anticorpo lgG2c no soro. Os soros de camun-dongos (n = 3 a 6) imunizados com CpG, 3 antígenos Mtb Rv diferentes, ou uma combinação dos mesmos são coletados 1 semana depois da 3a imunização e testa-dos para o antígeno específico para anticorpos lgG2c por ELISA. Os soros de grupos CpG são testados contra todos os antígenos Rv, enquanto outros soros são testados contra o antígeno Rv usado para a imunização. Os dados mostrados são a média ± SD (desvio padrão) de um experimento representativo. A figura 3C mostra o IFN-y liberado por esplenócitos estimulados por antígenos. Os esplenócitos de ca-mundongos imunizados com CpG, 3 antígenos Mtb Rv diferentes, ou uma combina-ção dos mesmos são coletados 3 semanas depois da 3a imunização e testado para o antígeno específico para a respostas da citoquina IFN-y por ELISA. Os esplenócitos são incubados por 72 h em um meio composto, ou em 10 pg/mL de antígenos Rv usados para a imunização. Os dados mostrados são a média ± SD (desvio padrão) (n = 3) em um experimento representativo. A figura 3a mostra frequências relativas de Esplenócitos TNF+ em resposta ao estímulo específico por antígeno. Os esplenócitos de camundongos imunizados com CpG, 3 antígenos Mtb Rv diferentes, ou uma combinação dos mesmos são coletados 3 semanas depois da 3a imunização e tes-tado para respostas da citoquina TNF específicas para o antígeno por ELISPOT. Os esplenócitos são incubados por 48 h em um meio composto, ou em 10 pg/mL dos antígenos Rv usados para a imunização. Os dados mostrados são a média ± SD (desvio padrão) (n = 3) em um experimento representativo.
[0053]A figura 4A-4B mostra a imunogenicidade de proteínas de fusão ID83 e ID93 com o GLA-SE em camundongos C57BL/6. A figura 4A mostra os pontos fi-nais de titulação para os anticorpos lgG1 e lgG2 no soro específicos para o antígeno. Os soros de camundongos (n = 3 a 6) imunizados com salina, ID83, ou proteína de fusão ID93 em formulações de adjuvante de GLA-SE são coletados 1 semana depois da 3a imunização e testado para anticorpos lgG1 e lgG2c específicos para ID83 e ID93 por ELISA. Os dados mostrados são a média ± SD (desvio padrão) em um experimento representativo. A figura 4B mostra que os níveis de IFN-y liberado pelo antígeno estimularam os esplenócitos. Os esplenócitos de camundongos imunizados com ID83 ou ID93 na formulação de adjuvante de GLA-SE são coletados 3 semanas depois da 3a imunização e testados para o antígeno específico para a respostas da citoquina IFN-y por ELISA. Os esplenócitos são incubados por 72 h em um meio composto de 3 pg/mL ConA, ou 10 pg/mL de proteínas de fusão ID83 ou ID93. Os dados mostrados são a média ± SD (desvio padrão) (n = 3) em um experimento representativo.
[0054]As figuras 5A-5B mostram a imunogenicidade de ID83 com formula-ções de adjuvante diferentes em camundongos C57BL/6. A figura 5A mostra os pon-tos finais de titulação para os anticorpos lgG1 e lgG2 no soro específicos para o an-tígeno. Os soros de camundongos (n = 3 a 6) imunizado com a salina, ou proteína de fusão ID83 com formulações de adjuvante diferentes são coletados 1 semana depois da 3a imunização e testado para Anticorpos lgG1 e lgG2c específicos para ID83 por ELISA. Os dados mostrados são a média ± SD (desvio padrão) em um experimento representativo. A figura 5B mostra que os níveis de IFN-y liberado esplenócitos estimulados por antigenos. Os esplenócitos de camundongos imunizados com a salina ou ID83 com a formulação de adjuvante diferente são coletados 3 semanas depois da 3a imunização e testado para o antígeno específico para as respostas da citoquina IFN-y por ELISA. Os esplenócitos são incubados por 72 h em um meio composto de 3 pg/mL ConA, ou 10 pg/mL de proteínas de fusão ID83. Os dados mostrados são a média ± SD (desvio padrão) (n = 3) em um experimento re-presentativo.
[0055]A figura 6 mostra a sobrevivência depois da infecção com Mtb de por- cos-da-índia imunizados com a proteína de fusão ID83 com GLA/CpG-SE. Os por- cos-da-índia são imunizado com 1 dose de BCG, ou 3 doses de ID83 com Adjuvante GLA/CpG-SE, e provocado com um aerossol de dose baixa de M. tuberculosis H37Rv, 4 semanas depois do último estímulo. A sobrevivência foi monitorada por 200 dias até que 3/4 dos animais no grupo de placebo (salina) morressem.
[0056]As figuras 7A-7B mostram respostas imunes específicas para Ad5- ID83 e a proteção contra um teste de provocação com M. tuberculosis. A figura 7A mostra frequências relativas de IFN-y + esplenócitos em resposta ao estímulo espe-cífico por antígeno. Os esplenócitos de camundongos imunizados com a salina, ou 5 x 109 partículas virais Ad5-ID83 são coletados 3 semanas depois da 3a imunização e testados para o antígeno específico para a respostas da citoquina IFN-y por ELISPOT. Os esplenócitos são incubados por 48 h em um meio composto, ou em 10 pg/mL proteína de fusão de ID83. Os dados mostrados são a média ± SD (desvio padrão) (n = 3) em um experimento representativo. A figura 7B mostra Log10 CFU no pulmão de camundongos imunizados depois de um teste de provocação com ae-rossol com M. tuberculosis. Os pulmões de camundongos (n = 7) imunizado com a salina, ou 5 x 109 partículas virais Ad5-ID83 são coletados 4 semanas depois de um teste de provocação com aerossol com 50 a 100 bacilos de Mtb. As CFU foram con-tadas depois de 2 semanas de crescimento in vitro na placa de ágar-ágar. Os dados mostrados são a média ± média do erro padrão, de um experimento representativo.
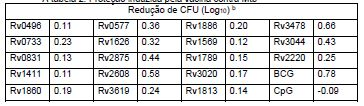
[0057]A figura 8 mostra que a sobrevivência camundongos SWR infectados com M. tuberculosis (n = 8) tratado com uma combinação de antibióticos (Rx; rifam- pina+ioniazida por 60 dias) + imunoterapia (três injeções de uma mistura contendo Rv2608, Rv1813, e Rv3620 com GLA-SE), antibióticos sozinhos (Rx; rifampi- na+ioniazida por 60 dias), ou deixados não tratados (salina). Os resultados demons-tram que a combinação de drogas + imunoterapia estende a sobrevivência de ca-mundongos infectados com M. tuberculosis.
[0058]A figura 9 mostra os resultados de experimentos de ELISA nos quais uma lista com saliva positiva, amostras de soro com Tb confirmada (n = 80 a 92) e uma lista com Tb negativa, e soro de controle saudável (n = 40 a 46) são analisados para a reatividade com antígenos Tb selecionados. Os resultados demonstram que 100 % de respostas positivas podem ser obtidas empregando combinações de antí-genos diferentes.
BREVE DESCRIÇÃO DOS IDENTIFICADORES DE sequência.
[0059]SEQ ID NO: 1 representa a sequência de aminoácidos predita para a Mtb Rv0164.
[0060]SEQ ID NO: 2 representa a sequência de uma sequência de ácido nu- cléico amplificada por PCR que codifica a Mtb Rv0164.
[0061]SEQ ID NO: 3 representa a sequência de aminoácidos de uma Mtb Rv0164 recombinante, incluindo a His marcada.
[0062]SEQ ID NO: 4 e 5 representa iniciadores usados para amplificar a Mtb Rv0164.
[0063]SEQ ID NO: 6 representa a sequência de aminoácidos predita para a Mtb Rv0496.
[0064]SEQ ID NO: 7 representa a sequência de uma sequência de ácido nu- cléico amplificada por PCR que codifica a Mtb Rv0496.
[0065]SEQ ID NO: 8 representa a sequência de aminoácidos de uma Mtb Rv0496 recombinante, incluindo a His marcada.
[0066]SEQ ID NO: 9 e 10 representam iniciadores usados para amplificar a Mtb Rv0496.
[0067]SEQ ID NO: 11 representa a sequência de aminoácidos predita para a Mtb Rv1738.
[0068]SEQ ID NO: 12 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Mtb Rv1738.
[0069]SEQ ID NO: 13 representa a sequência de aminoácidos de uma Mtb Rv1738 recombinante, incluindo a His marcada.
[0070]SEQ ID NO: 14 e 15 representam iniciadores usados para amplificar a Mtb Rv1738.
[0071]SEQ ID NO: 16 representa a sequência de aminoácidos predita para a Mtb Rv1813.
[0072]SEQ ID NO: 17 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Mtb Rv1813.
[0073]SEQ ID NO: 18 representa a sequência de aminoácidos de uma Mtb Rv1813 recombinante, incluindo a His marcada.
[0074]SEQ ID NO: 19 e 20 representam iniciadores usados para amplificar a Mtb Rv1813.
[0075]SEQ ID NO: 21 representa a sequência de aminoácidos predita para a Mtb Rv2389.
[0076]SEQ ID NO: 22 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Mtb Rv2389.
[0077]SEQ ID NO: 23 representa a sequência de aminoácidos de uma Mtb Rv2389 recombinante, incluindo a His marcada.
[0078]SEQ ID NO: 24 e 25 representam iniciadores usados para amplificar a Mtb Rv2389.
[0079]SEQ ID NO: 26 representa a sequência de aminoácidos predita para a Mtb Rv2608. SEQ ID NO: 27 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Mtb Rv2608.
[0080]SEQ ID NO: 28 representa a sequência de aminoácidos de uma Mtb Rv2608 recombinante, incluindo a His marcada.
[0081]SEQ ID NO: 29 e 30 representam iniciadores usados para amplificar a Mtb Rv2608.
[0082]SEQ ID NO: 31 representa a sequência de aminoácidos predita para a Mtb Rv2866.
[0083]SEQ ID NO: 32 e 33 representam iniciadores usados para amplificar a Mtb Rv2866.
[0084]SEQ ID NO: 34 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Mtb Rv2866.
[0085]SEQ ID NO: 35 representa a sequência de aminoácidos de uma Mtb Rv2866 recombinante, incluindo a His marcada.
[0086]SEQ ID NO: 36 representa a sequência de aminoácidos predita para a Mtb Rv3020.
[0087]SEQ ID NO: 37 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Mtb Rv3020.
[0088]SEQ ID NO: 38 representa a sequência de aminoácidos de uma Mtb Rv3020 recombinante, incluindo a His marcada.
[0089]SEQ ID NO: 39 e 40 representam iniciadores usados para amplificar a Mtb Rv3020.
[0090]SEQ ID NO: 41 representa a sequência de aminoácidos predita para a Mtb Rv3478.
[0091]SEQ ID NO: 42 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Mtb Rv3478.
[0092]SEQ ID NO: 43 representa a sequência de aminoácidos de uma Mtb Rv3478 recombinante, incluindo a His marcada.
[0093]SEQ ID NO: 44 e 45 representam iniciadores usados para amplificar a Mtb Rv3478.
[0094]SEQ ID NO: 46 representa a sequência de aminoácidos predita para a Mtb Rv3619.
[0095]SEQ ID NO: 47 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Mtb Rv3619.
[0096]SEQ ID NO: 48 representa a sequência de aminoácidos de uma Mtb Rv3619 recombinante, incluindo a His marcada.
[0097]SEQ ID NO: 49 e 50 representam iniciadores usados para amplificar a Mtb Rv3619.
[0098]SEQ ID NO: 51 representa a sequência de aminoácidos predita para a Mtb Rv3620.
[0099]SEQ ID NO: 52 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Mtb Rv3620.
[00100]SEQ ID NO: 53 representa a sequência de aminoácidos de uma Mtb Rv3620 recombinante, incluindo a His marcada.
[00101]SEQ ID NO: 54 e 55 representam iniciadores usados para amplificar a Mtb Rv3620.
[00102]SEQ ID NO: 56 representa a sequência de aminoácidos predita para a Mtb Rv3810.
[00103]SEQ ID NO: 57 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Mtb Rv3810.
[00104]SEQ ID NO: 58 representa a sequência de aminoácidos de uma Mtb Rv3810 recombinante, incluindo a His marcada.
[00105]SEQ ID NO: 59 e 60 representam iniciadores usados para amplificar a Mtb Rv3810.
[00106]SEQ ID NO: 61 representa a sequência de aminoácidos predita para a Mtb Rv3876.
[00107]SEQ ID NO: 62 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Mtb Rv3876.
[00108]SEQ ID NO: 63 representa a sequência de aminoácidos de uma Mtb Rv3876 recombinante, incluindo a His marcada.
[00109]SEQ ID NO: 64 e 65 representam iniciadores usados para amplificar a Mtb Rv3876.
[00110]SEQ ID NO: 66 representa uma sequência de polinucleotídeo que codifica o polipeptídeo de fusão Mtb36f.1.
[00111]SEQ ID NO: 67 representa a sequência de aminoácidos do polipeptí-deo de fusão Mtb Mtb36f.1 recombinante, incluindo a His marcada..
[00112]SEQ ID NO: 68-71 representam iniciadores usados na amplificação e na clonagem da Mtb36f.1.
[00113]SEQ ID NO: 72 representa uma sequência de polinucleotídeo que codifica o polipeptídeo de fusão ID58.
[00114]SEQ ID NO: 73 a 78 representam iniciadores usados na amplificação e na clonagem da ID58.
[00115]SEQ ID NO: 79 representa a sequência de aminoácidos do polipeptí-deo de fusão Mtb ID58, incluindo a His marcada.
[00116]SEQ ID NO: 80 representa uma sequência de polinucleotídeo que codifica o polipeptídeo de fusão ID69.
[00117]SEQ ID NO: 81 e 82 representam iniciadores usados na amplificação e na clonagem da ID69.
[00118]SEQ ID NO: 83 representa a sequência de aminoácidos do polipeptí-deo de fusão Mtb ID69 recombinante, incluindo a His marcada.
[00119]SEQ ID NO: 84 representa uma sequência de polinucleotídeo que codifica o polipeptídeo de fusão ID83.
[00120]SEQ ID NO: 85 a 90 representam iniciadores usados na amplificação e na clonagem da ID83.
[00121]SEQ ID NO: 91 representa a sequência de aminoácidos do polipeptí-deo de fusão Mtb ID83 recombinante, incluindo a His marcada.
[00122]SEQ ID NO: 92 representa uma sequência de polinucleotídeo que codifica o polipeptídeo de fusão ID94.
[00123]SEQ ID NO: 93 e 94 representam iniciadores usados na amplificação e na clonagem da ID94.
[00124]SEQ ID NO: 95 representa a sequência de aminoácidos do polipeptí-deo de fusão Mtb ID94 recombinante, incluindo a His marcada.
[00125]SEQ ID NO: 96 representa uma sequência de polinucleotídeo que codifica o polipeptídeo de fusão ID95.
[00126]SEQ ID NO: 97 representa a sequência de aminoácidos do polipeptí-deo de fusão Mtb ID95 recombinante, incluindo a His marcada.
[00127]SEQ ID NO: 98 representa uma sequência de polinucleotídeo que codifica o polipeptídeo de fusão ID 120.
[00128]SEQ ID NO: 99 representa a sequência de aminoácidos do polipeptí-deo de fusão Mtb ID120 recombinante, incluindo a His marcada.
[00129]SEQ ID NO: 100 representa a sequência de aminoácidos predita para a Rv0054.
[00130]SEQ ID NO: 101 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv0054.
[00131]SEQ ID NO: 102 representa a sequência de aminoácidos de uma Rv0054 recombinante, incluindo a His marcada.
[00132]SEQ ID NO: 103 representa a sequência de aminoácidos predita para a Rv0164.
[00133]SEQ ID NO: 104 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv0164.
[00134]SEQ ID NO: 105 representa a sequência de aminoácidos de uma Rv0164 recombinante, incluindo a His marcada.
[00135]SEQ ID NO: 106 representa a sequência de aminoácidos predita para a Rv0410.
[00136]SEQ ID NO: 107 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv0410.
[00137]SEQ ID NO: 108 representa a sequência de aminoácidos de uma Rv0410 recombinante, incluindo a His marcada.
[00138]SEQ ID NO: 109 representa a sequência de aminoácidos predita para a Rv0496.
[00139]SEQ ID NO: 110 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv0496.
[00140]SEQ ID NO: 111 representa a sequência de aminoácidos de uma Rv0496 recombinante, incluindo a His marcada.
[00141]SEQ ID NO: 112 representa a sequência de aminoácidos predita para a Rv0655.
[00142]SEQ ID NO: 113 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv0655.
[00143]SEQ ID NO: 114 representa a sequência de aminoácidos de uma Rv0655 recombinante, incluindo a His marcada.
[00144]SEQ ID NO: 115 representa a sequência de aminoácidos predita para a Rv0831.
[00145]SEQ ID NO: 116 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv0831.
[00146]SEQ ID NO: 117 representa a sequência de aminoácidos de uma Rv0831 recombinante, incluindo a His marcada.
[00147]SEQ ID NO: 118 representa a sequência de aminoácidos predita para a Rv1009.
[00148]SEQ ID NO: 119 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv1009.
[00149]SEQ ID NO: 120 representa a sequência de aminoácidos de uma Rv1009 recombinante, incluindo a His marcada.
[00150]SEQ ID NO: 121 representa a sequência de aminoácidos predita para a Rv1099.
[00151]SEQ ID NO: 122 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv1099.
[00152]SEQ ID NO: 123 representa a sequência de aminoácidos de uma Rv1099 recombinante, incluindo a His marcada.
[00153]SEQ ID NO: 124 representa a sequência de aminoácidos predita para a Rv1240.
[00154]SEQ ID NO: 125 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv1240.
[00155]SEQ ID NO: 126 representa a sequência de aminoácidos de uma Rv1240 recombinante, incluindo a His marcada.
[00156]SEQ ID NO: 127 representa a sequência de aminoácidos predita para a Rv1288.
[00157]SEQ ID NO: 128 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv1288.
[00158]SEQ ID NO: 129 representa a sequência de aminoácidos de uma Rv1288 recombinante, incluindo a His marcada.
[00159]SEQ ID NO: 130 representa a sequência de aminoácidos predita para a Rv1410.
[00160]SEQ ID NO: 131 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv1410.
[00161]SEQ ID NO: 132 representa a sequência de aminoácidos de uma Rv1410 recombinante, incluindo a His marcada.
[00162]SEQ ID NO: 133 representa a sequência de aminoácidos predita para a Rv1569.
[00163]SEQ ID NO: 134 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv1569.
[00164]SEQ ID NO: 135 representa a sequência de aminoácidos de uma Rv1569 recombinante, incluindo a His marcada.
[00165]SEQ ID NO: 136 representa a sequência de aminoácidos predita para a Rv1789.
[00166]SEQ ID NO: 137 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv1789.
[00167]SEQ ID NO: 138 representa a sequência de aminoácidos de uma Rv1789 recombinante, incluindo a His marcada.
[00168]SEQ ID NO: 139 representa a sequência de aminoácidos predita para a Rv1818.
[00169]SEQ ID NO: 140 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv1818.
[00170]SEQ ID NO: 141 representa a sequência de aminoácidos de uma Rv1818 recombinante, incluindo a His marcada.
[00171]SEQ ID NO: 142 representa a sequência de aminoácidos predita para a Rv1860.
[00172]SEQ ID NO: 143 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv1860.
[00173]SEQ ID NO: 144 representa a sequência de aminoácidos de uma Rv1860 recombinante, incluindo a His marcada.
[00174]SEQ ID NO: 145 representa a sequência de aminoácidos predita para a Rv1886.
[00175]SEQ ID NO: 146 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv1886.
[00176]SEQ ID NO: 147 representa a sequência de aminoácidos de uma Rv1886 recombinante, incluindo a His marcada.
[00177]SEQ ID NO: 148 representa a sequência de aminoácidos predita para a Rv1908.
[00178]SEQ ID NO: 149 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv1908.
[00179]SEQ ID NO: 150 representa a sequência de aminoácidos de uma Rv1908 recombinante, incluindo a His marcada.
[00180]SEQ ID NO: 151 representa a sequência de aminoácidos predita para a Rv2032.
[00181]SEQ ID NO: 152 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv2032.
[00182]SEQ ID NO: 153 representa a sequência de aminoácidos de uma Rv2032 recombinante, incluindo a His marcada.
[00183]SEQ ID NO: 154 representa a sequência de aminoácidos predita para a Rv2220.
[00184]SEQ ID NO: 155 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv2220.
[00185]SEQ ID NO: 156 representa a sequência de aminoácidos de uma Rv2220 recombinante, incluindo a His marcada.
[00186]SEQ ID NO: 157 representa a sequência de aminoácidos predita para a Rv2608.
[00187]SEQ ID NO: 158 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv2608.
[00188]SEQ ID NO: 159 representa a sequência de aminoácidos de uma Rv2608 recombinante, incluindo a His marcada.
[00189]SEQ ID NO: 160 representa a sequência de aminoácidos predita para a Rv2623.
[00190]SEQ ID NO: 161 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv2623.
[00191]SEQ ID NO: 162 representa a sequência de aminoácidos de uma Rv2623 recombinante, incluindo a His marcada.
[00192]SEQ ID NO: 163 representa a sequência de aminoácidos predita para a Rv2875.
[00193]SEQ ID NO: 164 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv2875.
[00194]SEQ ID NO: 165 representa a sequência de aminoácidos de uma Rv2875 recombinante, incluindo a His marcada.
[00195]SEQ ID NO: 166 representa a sequência de aminoácidos predita para a Rv3044.
[00196]SEQ ID NO: 167 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv3044.
[00197]SEQ ID NO: 168 representa a sequência de aminoácidos de uma Rv3004 recombinante, incluindo a His marcada.
[00198]SEQ ID NO: 169 representa a sequência de aminoácidos predita para a Rv3310.
[00199]SEQ ID NO: 170 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv3310.
[00200]SEQ ID NO: 171 representa a sequência de aminoácidos de uma Rv3310 recombinante, incluindo a His marcada.
[00201]SEQ ID NO: 172 representa a sequência de aminoácidos predita pa-ra a Rv3619.
[00202]SEQ ID NO: 173 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv3619.
[00203]SEQ ID NO: 174 representa a sequência de aminoácidos de uma Rv3619 recombinante, incluindo a His marcada.
[00204]SEQ ID NO: 175 representa a sequência de aminoácidos predita para a Rv3810.
[00205]SEQ ID NO: 176 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv3810.
[00206]SEQ ID NO: 177 representa a sequência de aminoácidos de uma Rv3810 recombinante, incluindo a His marcada.
[00207]SEQ ID NO: 178 representa a sequência de aminoácidos predita para a Rv3881.
[00208]SEQ ID NO: 179 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv3881.
[00209]SEQ ID NO: 180 representa a sequência de aminoácidos de uma Rv3881 recombinante, incluindo a His marcada.
[00210]SEQ ID NO: 181 representa a sequência de aminoácidos predita para a Rv0455.
[00211]SEQ ID NO: 182 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv0455.
[00212]SEQ ID NO: 183 representa a sequência de aminoácidos de uma Rv0455 recombinante, incluindo a His marcada.
[00213]SEQ ID NO: 184 representa a sequência de aminoácidos predita para a Rv0577.
[00214]SEQ ID NO: 185 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv0577.
[00215]SEQ ID NO: 186 representa a sequência de aminoácidos de uma Rv0577 recombinante, incluindo a His marcada.
[00216]SEQ ID NO: 187 representa a sequência de aminoácidos predita para a Rv1626.
[00217]SEQ ID NO: 188 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv1626.
[00218]SEQ ID NO: 189 representa a sequência de aminoácidos de uma Rv1626 recombinante, incluindo a His marcada.
[00219]SEQ ID NO: 190 representa a sequência de aminoácidos predita para a Rv0733.
[00220]SEQ ID NO: 191 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv0733.
[00221]SEQ ID NO: 192 representa a sequência de aminoácidos de uma Rv0733 recombinante, incluindo a His marcada.
[00222]SEQ ID NO: 193 representa a sequência de aminoácidos predita para a Rv2520.
[00223]SEQ ID NO: 194 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv2520.
[00224]SEQ ID NO: 195 representa a sequência de aminoácidos de uma Rv2520 recombinante, incluindo a His marcada.
[00225]SEQ ID NO: 196 representa a sequência de aminoácidos predita para a Rv1253.
[00226]SEQ ID NO: 197 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv1253.
[00227]SEQ ID NO: 198 representa a sequência de aminoácidos de uma Rv1253 recombinante, incluindo a His marcada.
[00228]SEQ ID NO: 199 representa a sequência de aminoácidos predita para a Rv1980.
[00229]SEQ ID NO: 200 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv1980.
[00230]SEQ ID NO: 201 representa a sequência de aminoácidos de uma Rv1980 recombinante, incluindo a His marcada.
[00231]SEQ ID NO: 202 representa a sequência de aminoácidos predita para a Rv3628.
[00232]SEQ ID NO: 203 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv3628.
[00233]SEQ ID NO: 204 representa a sequência de aminoácidos de uma Rv3628 recombinante, incluindo a His marcada.
[00234]SEQ ID NO: 205 representa a sequência de aminoácidos predita para a Rv1884.
[00235]SEQ ID NO: 206 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv1884.
[00236]SEQ ID NO: 207 representa a sequência de aminoácidos de uma Rv1884 recombinante, incluindo a His marcada.
[00237]SEQ ID NO: 208 representa a sequência de aminoácidos predita para a Rv3872.
[00238]SEQ ID NO: 209 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv3872.
[00239]SEQ ID NO: 210 representa a sequência de aminoácidos de uma Rv3872 recombinante, incluindo a His marcada.
[00240]SEQ ID NO: 211 representa a sequência de aminoácidos predita para a Rv3873.
[00241]SEQ ID NO: 212 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv3873.
[00242]SEQ ID NO: 213 representa a sequência de aminoácidos de uma Rv3873 recombinante, incluindo a His marcada.
[00243]SEQ ID NO: 214 representa a sequência de aminoácidos predita para a Rv1511.
[00244]SEQ ID NO: 215 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv1511.
[00245]SEQ ID NO: 216 representa a sequência de aminoácidos de uma Rv1511 recombinante, incluindo a His marcada.
[00246]SEQ ID NO: 217 representa uma sequência de polinucleotídeo que codifica o polipeptídeo de fusão ID93.
[00247]SEQ ID NO: 218 a 225 representam iniciadores usados na amplifica-ção e na clonagem da ID93.
[00248]SEQ ID NO: 226 representa a sequência de aminoácidos do polipep-tídeo de fusão Mtb ID93 recombinante, incluindo a His marcada.
[00249]SEQ ID NO: 227 representa uma sequência de polinucleotídeo que codifica o polipeptídeo de fusão ID91.
[00250]SEQ ID NO: 228 a 235 representa iniciadores usados na amplificação e na clonagem da ID91.
[00251]SEQ ID NO: 236 representa a sequência de aminoácidos do polipep-tídeo de fusão Mtb ID91 recombinante, incluindo a His marcada.
[00252]SEQ ID NO: 237 representa uma sequência de polinucleotídeo que codifica o polipeptídeo de fusão ID71.
[00253]SEQ ID NO: 238 a 244 representa iniciadores usados na amplificação e na clonagem da ID71.
[00254]SEQ ID NO: 245 representa a sequência de aminoácidos do polipep-tídeo de fusão Mtb ID71 recombinante, incluindo a His marcada.
[00255]SEQ ID NO: 246 representa uma sequência de polinucleotídeo que codifica o polipeptídeo de fusão ID 114.
[00256]SEQ ID NO: 247 a 250 representa iniciadores usados na amplificação e na clonagem da ID114.
[00257]SEQ ID NO: 251 representa a sequência de aminoácidos do polipep-tídeo de fusão Mtb ID114 recombinante, incluindo a His marcada.
[00258]SEQ ID NO: 252 representa uma sequência de polinucleotídeo que codifica o polipeptídeo de fusão ID 125.
[00259]SEQ ID NO: 253 a 256 representa iniciadores usados na amplificação e na clonagem da ID125.
[00260]SEQ ID NO: 257 representa a sequência de aminoácidos do polipep-tídeo de fusão Mtb ID125 recombinante, incluindo a His marcada.
[00261]SEQ ID NO: 258 representa uma sequência de polinucleotídeo que codifica o polipeptídeo de fusão DID85.
[00262]SEQ ID NO: 259 a 264 representa iniciadores usados na amplifica-ção e na clonagem da DID85.
[00263]SEQ ID NO: 265 representa a sequência de aminoácidos do polipep-tídeo de fusão Mtb DID85 recombinante, incluindo a His marcada.
[00264]SEQ ID NO: 266 representa uma sequência de polinucleotídeo que codifica o polipeptídeo de fusão DID92.
[00265]SEQ ID NO: 267 a 272 representa iniciadores usados na amplificação e na clonagem da DID92.
[00266]SEQ ID NO: 273 representa a sequência de aminoácidos do polipep-tídeo de fusão Mtb DID92 recombinante, incluindo a His marcada.
[00267]SEQ ID NO: 274 representa uma sequência de polinucleotídeo que codifica o polipeptídeo de fusão DID108.
[00268]SEQ ID NO: 275 a 282 representa iniciadores usados na amplificação e na clonagem da DID108.
[00269]SEQ ID NO: 283 representa a sequência de aminoácidos do polipep-tídeo de fusão Mtb DID108 recombinante, incluindo a His marcada.
[00270]SEQ ID NO: 284 representa uma sequência de polinucleotídeo que codifica o polipeptídeo de fusão DID93.
[00271]SEQ ID NO: 285 a 290 representa iniciadores usados na amplifica- ção e na clonagem da DID93.
[00272]SEQ ID NO: 291 representa a sequência de aminoácidos do polipep-tídeo de fusão Mtb DID93 recombinante, incluindo a His marcada.
[00273]SEQ ID NO: 292 representa a sequência de aminoácidos predita para a Rv3875.
[00274]SEQ ID NO: 293 representa a sequência de uma sequência de ácido nucléico amplificada por PCR que codifica a Rv3875.
[00275]SEQ ID NO: 294 representa a sequência de aminoácidos de uma Rv3875 recombinante, incluindo a His marcada.
[00276]SEQ ID NO: 295 e 296 representa iniciadores usados na amplificação e na clonagem da Rv0577.
[00277]SEQ ID NO: 297 e 298 representa iniciadores usados na amplificação e na clonagem da Rv1626.
[00278]SEQ ID NO: 299-300 representa iniciadores usados na amplificação e na clonagem da Rv0733.
[00279]SEQ ID NO: 301-302 representa iniciadores usados na amplificação e na clonagem da Rv2520.
[00280]SEQ ID NO: 303-304 representa iniciadores usados na amplificação e na clonagem da Rv1253.
[00281]SEQ ID NO: 305-306 representa iniciadores usados na amplificação e na clonagem da Rv1980.
[00282]SEQ ID NO: 307-308 representa iniciadores usados na amplificação e na clonagem da Rv3628.
[00283]SEQ ID NO: 309-310 representa iniciadores usados na amplificação e na clonagem da Rv1844.
[00284]SEQ ID NO: 311-312 representa iniciadores usados na amplificação e na clonagem da Rv3872.
[00285]SEQ ID NO: 313-314 representa iniciadores usados na amplificação e na clonagem da Rv3873.
[00286]SEQ ID NO: 315-316 representa iniciadores usados na amplificação e na clonagem da Rv1511.
[00287]SEQ ID NO: 317-318 representa iniciadores usados na amplificação e na clonagem da Rv3875.
DESCRIÇÃO DETALHADA.
[00288]A presente invenção se refere a composições altamente antigêni- cas/imunogênicas compreendendo antígenos de Mycobacterium. As composições da presente invenção geralmente compreendem pelo menos dois polipeptídeos he- terólogos da espécie Mycobacterium do complexo de tuberculose. Uma espécie Mycobacterium do complexo de tuberculose inclui aquelas espécies tradicionalmente consideradas como a causa da doença tuberculose, bem como espécies de Myco-bacterium ambientais e oportunistas que causam a tuberculose e a doença do pul-mão em pacientes imunocomprometidos, tais como pacientes com a Aids, por exemplo, Mycobacterium tuberculosis (Mtb), Mycobacterium bovis, ou Mycobacterium africanum, BCG, Mycobacterium avium, Mycobacterium intracellulare, Mycobacterium celatum, Mycobacterium genavense, Mycobacterium haemophilum, Mycobacterium kansasii, Mycobacterium simiae, Mycobacterium vaccae, Mycobacterium for- tuitum, e Mycobacterium scrofulaceum (ver, por exemplo, Harrison's Prionci pies of Internal Medicine, volume 1, páginas 1004-1014 e 1019-1020. Em uma modalidade preferida, as espécies de Mycobacterium a ser evitadas, tratadas ou diagnosticadas de acordo com a invenção é a Mycobacterium tuberculosis (Mtb). As sequências de antígenos das espécies de Mycobacterium estão prontamente disponíveis. Por exemplo, as sequências de Mycobacterium tuberculosis podem ser encontradas em Cole et al., Nature 393:537 (1998) e pode ser encontrada em páginas da web, tais como os mantidos pela Wellcome Trust Sanger Institute e Institut Pasteur.
A. Antígenos de Mycobacterium e Fusões dos Mesmos.
[00289]A presente invenção, em um aspecto, fornece polipeptídeos isolados de Mycobacterium, tal como aqui descritos, incluindo polipeptídeos de fusão, e com-posições contendo os mesmos. Geralmente, um polipeptídeo de acordo com a in-venção será um polipeptídeo isolado e pode ser um fragmento (por exemplo, uma porção antigênica/imunogênica) de uma sequência de aminoácidos aqui divulgada, ou pode compreender uma sequência inteira de aminoácidos aqui divulgada. Os po-lipeptídeos, fragmentos antigênicos/imunogênicos, e outras variantes dos mesmos, de acordo com a invenção, podem ser preparados técnicas convencionais recomb- ninantes e/ou sintéticas.
[00290]Em certas modalidades preferidas, os polipeptídeos de acordo com a invenção são antigênicos/imunogênicos, isto é, eles reagem detectavelmente dentro de um imunoensaio (tal como um ELISA ou ensaio de estimulação de célula T) com antisoros e/ou células T de um paciente infectado. A avaliação da atividade imuno- gênica pode ser realizada usando técnicas bem conhecidas do técnico versado. Por exemplo, tais avaliações podem ser realizadas usando métodos, tais como os des-critos em Harlow e Lane, Antibodies: A Laboratory Manual, Cold Spring Harbor Labo-ratory, 1988. Em um exemplo ilustrativo, um polipeptídeo pode ser imobilizado sobre um suporte sólido e colocado em contato com os soros de pacientes para permitir a ligação dos anticorpos dentro dos soros ao polipeptídeo imobilizado. Os soros não ligados podem então ser removidos e os anticorpos ligados são detectados utilizan-do, por exemplo, proteína A marcada com 125l.
[00291]Como seria reconhecido por um técnico versado, as porções imuno- gênicas dos polipeptídeos aqui divulgados são também abrangidas pela presente invenção. Uma “porção imunogênica”, tal como aqui utilizado, é um fragmento de um polipeptídeo imunogênico de acordo com a invenção que é, ele mesmo, imunologi- camente reativo (isto é, especificamente se liga) com com os receptores de antíge- nos nas superfícies das células B e/ou T que reconhecem o polipeptídeo. As porções imunogênicas podem ser geralmente identificadas usando técnicas bem conhecidas, tais como aquelas resumidas em Paul, Fundamental Immunology, 3a edição, 243-247 (Raven Press, 1993) e as referências citadas no mesmo. Tais técnicas incluem polipeptídeos de avaliação da capacidade de reagir com anticorpos específicos para o antígeno, antisoros e/ou linhagens de células T ou clones. Tal como aqui utilizado, os antisoros e os anticorpos são "específicos para o antígeno" se eles especificamente se ligarem a um antígeno (isto é, eles reagem com a proteína em um imunoensaio, e não reagirem detectavelmente com proteínas não relacionadas). Tais antisoros e os anticorpos podem ser preparados tal como aqui descrito, e usando técnicas bem conhecidas.
[00292]Em uma modalidade particular, uma porção antigênica/imunogênica de um polipeptídeo da presente invenção é uma porção que reage com antisoros e/ou células T a um nível que não é substancialmente menor do que a reatividade do polipeptídeo de corpo inteiro (por exemplo, em um e/ou ELISA T ensaio de reativida-de de célula). Preferivelmente, o nível da atividade imunogênica da porção antigêni-ca/imunogênica é pelo menos aproximadamente 50 %, preferivelmente pelo menos aproximadamente 70 % e bem mais preferivelmente maior do que aproximadamente 90 % da imunogenicidade do polipeptídeo de corpo inteiro. Em exemplos alguns, as porções imunogênicas preferidas serão identificadas possuindo um nível da ativida-de imunogênica maior do que aquela do polipeptídeo de corpo inteiro corresponden-te, por exemplo, tendo atividade imunogênica maior do que aproximadamente 100 % ou mais, ou de 150 %.
[00293]Uma composição de polipeptídeo de acordo com a invenção também pode compreender um ou mais polipeptídeos que são imunologicamente reativos com células T anticorpos de e/ou gerados contra um polipeptídeo de acordo com a invenção, particularmente um polipeptídeo que revela uma sequência de aminoáci- dos aqui, ou a um fragmento imunogênico ou variante do mesmo.
[00294]Em outra modalidade de acordo com a invenção, os polipeptídeos são contanto que compreenda um ou mais polipeptídeos que são capazes de eliciar células T anticorpos de e/ou que são imunologicamente reativos com um ou mais polipeptídeos aqui descritos, ou um ou mais polipeptídeos codificados por sequên-cias contíguas de polinucleotídeo contidas nas sequências de polinucleotídeo aqui divulgadas, ou fragmentos imunogênicos ou variantes dos mesmos, ou a uma ou mais sequências de polinucleotídeo que hibridizam uma ou mais destas sequências sob condições de severidade moderada à alta.
[00295]A presente invenção também fornece fragmentos de polipeptídeo, in-cluindo fragmentos antigênicos/imunogênicos, compreendendo pelo menos aproxi-madamente 5, 10, 15, 20, 25, 50, ou 100 aminoácidos contíguos, ou mais, incluindo todos os comprimentos intermediários, de uma composição de polipeptídeo aqui apresentada, ou aqueles codificados por uma sequência de polinucleotídeo aqui apresentada.
[00296]Em outro aspecto, a presente invenção fornece variantes das com-posições de polipeptídeo aqui descritas. As variantes de polipeptídeo geralmente abrangidas pela presente invenção exibirão tipicamente pelo menos aproximada-mente ou uma identidade de 70 %, 75 %, 80 %, 85 %, 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, ou mais de 99 % (determinado tal como descrito abaixo), ao longo dhis comprimento, em relação à uma sequência de polipeptídeo aqui apresentada.
[00297]Um polipeptídeo "variante", tal como o termo é aqui usado, é um po-lipeptídeo que tipicamente se diferencia de um polipeptídeo especificamente divul-gado aqui em uma ou mais substituições, eliminações, adições e/ou inserções. Tais variantes podem ser de ocorrência natural ou podem ser sinteticamente geradas, por exemplo, modificando uma ou mais das sequências acima, de um polipeptídeo de acordo com a invenção, e avaliando a sua atividade imunogênica tal como aqui des-crito, utilizando qualquer uma das diversas técnicas bem conhecidas na técnica.
[00298]Por exemplo, certas variantes ilustrativas dos polipeptídeos de acordo com a invenção incluem aquelas em que uma ou mais porções, tal como uma sequência líder de terminação N ou o domínio transmembrana, foram removidos. Outras variantes ilustrativas incluem variantes em que uma pequena porção (por exemplo, aproximadamente de 1 a 30 aminoácidos) foi removido do e/ou N- terminação C de uma proteína madura.
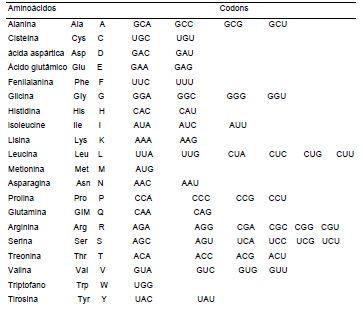
[00299]Em muitos exemplos, uma variante conterá substituições conserva-doras. Uma “substituição conservadora" é aquela na qual um aminoácido é substitu-ído por outro aminoácido que tem propriedades semelhantes, de tal modo que um técnico versado na técnica da química de peptídios esperaria que a estrutura secun-dária e natureza hidropática do polipeptídeo fosse substancialmente inalterada. Tal como descrito acima, as modificações podem ser feitas na estrutura dos polinucleo- tídeos e polipeptídeos da presente invenção e ainda obter uma molécula funcional que codifica variantes ou derivados de um polipeptídeo com características desejá-veis, por exemplo, com características imunogênicas. Quando é desejado alterar a sequência de aminoácidos de um polipeptídeo para criar um equivalente, ou até uma variante imunogênica melhorada, ou a porção de um polipeptídeo de acordo com a invenção, uma pessoa versada na técnica modificará tipicamente um ou mais dos codons da sequência de codificação de DNA de acordo com a Tabela 1.
[00300]Por exemplo, certos aminoácidos podem ser substituídos por outros aminoácidos em uma estrutura de proteína sem a perda apreciável da capacidade obrigatória interativa com estruturas tal como, por exemplo, regiões obrigatórias pelo antígeno de anticorpos ou sítios de ligação em moléculas de substrato. Como é a capacidade interativa e Nature de uma proteína que define a atividade funcional bio-lógica daquela proteína, certas substituições da sequência de aminoácidos podem ser feitas em uma sequência de proteína, e, naturalmente, a sua sequência de codi-ficação de DNA subjacente, e no entanto obter uma proteína com propriedades pa-recidas. É assim contemplado que as modificações diversas podem ser feitas nas sequências de peptídio das composições divulgadas, ou as sequências de DNA cor-respondentes codificando disseram peptídios sem a perda apreciável da sua utilida-de biológica ou atividade. TABELA 1.
[00301]Na criação de tais modificações, o índice hidropático de aminoácidos pode considerar-se. A importância do índice hidropático de aminoácido na conferi- ção de função biológica interativa sobre uma proteína é geralmente entendida na técnica (Kyte e Doolittle, 1982, incorporada aqui pela referência). Reconhece-se que o caráter hidropático relativo do aminoácido contribui para a estrutura secundária da proteína resultante, que por sua vez define a interação da proteína com outras moléculas, por exemplo, enzimas, substratos, receptores, DNA, anticorpos, antígenos, e assim por diante. Cada aminoácido foi destinou um índice hidropático com base na sua hidrofobicidade e características de carga (Kyte e Doolittle, 1982). Estes valores são: isoleucina (+4.5); valina (+4.2); leucina (+3.8); fenilalanina (+2.8); cystei- ne/cystine (+2.5); metionina (+1.9); alanina (+1.8); glicina (-0.4); treonina (-0.7); seri- na (-0.8); triptofano (-0.9); tirosina (-1.3); prolina (-1.6); histidina (-3.2); glutamato (- 3.5); glutamina (-3.5); aspartato (-3.5); asparagina (-3.5); lisina (-3.9); e arginina (- 4.5).
[00302]Conhece-se na técnica que certos aminoácidos podem ser substituídos por outros aminoácidos possuindo um índice hidropático semelhante ou resultado e ainda resultar em uma proteína com a atividade biológica semelhante, isto é ainda obter uma proteína biológica funcionalmente equivalente. Na criação de tais modificações, a substituição de aminoácidos cujos índices hidropático são dentro de ±2 é preferida, aqueles dentro de ±1 são particularmente preferidos, e aqueles dentro de ±0.5 são até mais particularmente preferidos. Também se entende na técnica que a substituição de aminoácidos parecidos pode ser feita eficazmente com base em hidrofilicidade.
[00303]Como detalhado em 4.554.101 Patentes dos Estados Unidos, o seguinte hidrofilicidade valoriza o foram destinado a resíduos de aminoácido: arginina (+3.0); lisina (+3.0); aspartato (+3.0 ± 1); glutamato (+3.0 ± 1); serina (+0.3); asparagina (+0.2); glutamina (+0.2); glicina (0); treonina (-0.4); prolina (-0.5 ± 1); alanina (- 0.5); histidina (-0.5); cisteína (-1.0); metionina (-1.3); valina (-1.5); leucina (-1.8); isoleucina (-1.8); tirosina (-2.3); fenilalanina (-2.5); triptofano (-3.4). Entende-se que um aminoácido pode ser substituído pelo outro que tem um hidrofilicidade semelhante valorizam e ainda obtêm um biologicamente equivalente, e especialmente, uma proteína imunologicamente equivalente. Em tais modificações, a substituição de aminoácidos cujos valores de hidrofilicidade são dentro de ±2 é preferida, aqueles dentro de ±1 são particularmente preferidos, e aqueles dentro de ±0.5 são até mais particularmente preferidos. Como delineado acima, as substituições de aminoácido são geralmente, desse modo, baseadas na semelhança relativa dos substituintes de cadeia do lado de aminoácido, por exemplo, a sua hidrofobicidade, hidrofilicidade, carga, tamanho, e assim por diante. As substituições exemplares que tomam diferentes das características precedentes na consideração são bem conhecidas daqueles com habilidade na técnica e incluem: arginina e lisina; glutamato e aspartato; serina e treonina; glutamina e asparagina; e valina, leucina e isoleucina.
[00304]Além disso, o polinucleotídeo qualquer pode ser também modificado para aumentar a estabilidade in vivo. As modificações possíveis incluem, mas são não limitadas a, a adição de flanquear sequências nas extremidades 5' e/ou 3'; o uso de phosphorothioate ou 2' O-methyl em vez de ligações phosphodiesterase na cadeia principal; e/ou a inclusão de bases não tradicionais, tais como inosine, queosine e wybutosine, bem como acetil-, metil-, tio- e outras formas modificadas de adenina, citidina, guanina, timina e uridine.
[00305]As substituições de aminoácido podem ser também feitas com base na semelhança em polaridade, carga, solubilidade, hidrofobicidade, hidrofilicidade e/ou natureza anfipática dos resíduos. Por exemplo, os aminoácidos negativamente carregados incluem o ácido aspártico e o ácido glutâmico; os aminoácidos positiva-mente carregados incluem a lisina e a arginina; e os aminoácidos com grupos dian-teiros polares não carregados possuindo valores de hidrofilicidade semelhantes in-cluem a leucina, a isoleucina e a valina; glicina e alanina; asparagina e glutamina; e serina, treonina, fenilalanina e tirosina. Outros grupos de aminoácidos que podem representar modificações conservadoras incluem: (1) ala, pro, gly, glu, asp, gim, asn, ser, thr; (2) cys, ser, tyr, thr; (3) vai, ile, leu, met, ala, phe; (4) lys, arg, his; e (5) phe, tyr, trp, his. Uma variante pode também, ou alternativamente, conter modificações não conservadoras. Em uma modalidade preferida, os polipeptídeos de variantes diferenciam-se de uma sequência nativa por substituição, eliminação ou adição de cinco aminoácidos ou menos.
[00306]As variantes podem também (ou alternativamente) ser modificadas por, por exemplo, a eliminação ou a adição de aminoácidos possuindo a influência mínima na imunogenicidade, estrutura secundária e natureza hidropático do polipeptídeo.
[00307]Como observado acima, os polipeptídeos podem compreender um sinal (ou líder) sequência na extremidade de terminação N da proteína, que co-de translaçãomente ou pós-de-translaçãomente dirige a transferência da proteína. O polipeptídeo também pode ser conjugado a um ligante ou outra sequência da facilidade de síntese, purificação ou identificação do polipeptídeo (por exemplo, poli-His), ou melhorar a ligação do polipeptídeo a um suporte sólido. Por exemplo, um polipeptídeo pode ser conjugado a uma região Fc da imunoglobulina.
[00308]Comparando sequências de polipeptídeo, duas sequências são disse ser "idêntico" se a sequência de aminoácidos nas duas sequências é o mesmo quando alinhado com a correspondência máxima, como descrito abaixo. As comparações entre duas sequências são tipicamente realizadas comparando as sequências por cima de uma janela de comparação para identificar e comparar regiões locais da semelhança de sequência. Uma “janela de comparação" tal como aqui utilizado, refere-se a um segmento de pelo menos aproximadamente 20 posições contíguas, normalmente de 30 a aproximadamente 75, de 40 a aproximadamente 50, no qual uma sequência pode ser comparada com uma sequência de referência do mesmo número de posições contíguas depois que as duas sequências forem otimamente alinhadas.
[00309]O ótimo alinhamento de sequências da comparação pode ser conduzido usando o programa Megalign na suite Lasergene do software bioinformatics (DNASTAR, Inc, Madison, Wl), usando parâmetros aleatórios. Este programa personifica vários esquemas de alinhamento descritos nas seguintes referências: dia hoff, M. O. (1978) um modelo da modificação evolutiva em proteínas - Matrizes para detectar relações distantes. Em dia hoff, M. O. (editor). Atlas da sequência de proteína e Estrutura, Fundação de Pesquisa Biomédica Nacional, volume de Washington, DC 5, Suppl. 3, páginas 345-358; Hein J. (1990) Aproximação Unificada de Alinhamento e páginas Phylogenes 626-645 Métodos em volume de Enzimologia 183, Academic Press, Inc, São Diego, Califórnia; Hlgdns, D. G. e Agudo, da tarde (1989) CABIOS 5:151-153; Myers, E.W. e Muller W. (1988) CABIOS 4:11-17; Robinson, E.D. (1971) Pente. Theor 77:105; Santou, N. Nes, M. (1987) Mol. Biol. Evol. 4:406-425; Sneath, P.H.A. e Sokal, R.R. (1973) Taxonomia Numérica - os Princípios e Prática de Taxo- nomia Numérica, Prensa de Homem livre, São Francisco, Califórnia; Wilbur, W.J. e Lipman, D.J.. (1983) Proc. Natl Acad., Sci. EUA 80:726-730.
[00310]Alternativamente, o ótimo alinhamento de sequências da comparação pode ser conduzido pelo algoritmo de identidade local de Smith e Waterman (1981) Add. APL. Matemática 2:482, pelo algoritmo de alinhamento de identidade de Needleman e Wunsch (1970) J. Mol. Biol. 48:443, pela pesquisa de métodos de semelhança de Pearson e Lipman (1988) Proc. Nat'l Acad. Sci. EUA 85: 2444, por implementações computadorizadas destes algoritmos (GAP, BESTFIT, BLAST, FASTA, e TFASTA no Pacote de Software de Genética de Wisconsin, Grupo de Computador de Genética (GCG), 575 Science doutor, Madison, Wl), ou por inspeção.
[00311]Um exemplo preferido de algoritmos que são adequados para determinar a identidade da sequência de por cento e a semelhança da sequência é a BLAST e BLAST 2.0 algoritmos, que são descritos em Altschul et al. (1977) Nucl. Ácidos Res. 25:3389-3402 e Altschul et al. (1990) J. Mol. Biol. 215:403-410, respec- tivameπte. BLAST e BLAST 2.0 podem ser usadas, por exemplo com os parâmetros aqui descritos, para determinar a identidade da sequência de por cento dos polinu- cleotídeos e polipeptídeos de acordo com a invenção. O software para executar análises de BLAST está publicamente disponível pelo Centro Nacional da Informação sobre Biotecnologia. Para sequências de aminoácido, uma matriz de marcação pode ser usada para calcular o resultado cumulativo. A extensão dos golpes de palavra em cada direção é parada quando: o resultado de alinhamento cumulativo cai pela quantidade X do seu valor máximo atingido; o resultado cumulativo vai ao zero ou abaixo, devido aos alinhamentos de resíduo de marcação negativa de um ou mais de acumulação; ou a extremidade de qualquer sequência é atingida. Os parâmetros de algoritmo de BLAST W, T e X determinam a sensibilidade e velocidade do alinhamento. Em uma aproximação preferida, "a percentagem da identidade de sequência" é determinada comparando duas sequências otimamente alinhadas por cima de uma janela da comparação de pelo menos 20 posições, em que a porção da sequência de polipeptídeo na janela de comparação pode compreender adições ou eliminações (isto é, gaps) de 20 por cento ou menos, normalmente 5 para 15 por cento, ou 10 para 12 por cento, comparando com as sequências de referência (que não compreende adições ou eliminações) para o ótimo alinhamento das duas sequências. A percentagem é calculada determinando o número de posições nas quais o resíduo de aminoácido idêntico ocorre em sequências de ambos para produzir o número de posições combinadas, dividindo o número de posições combinadas pelo número total de posições na sequência de referência (isto é, o tamanho de janela) e multiplicando os resultados por 100 para produzir a percentagem da identidade de sequência.
[00312]Em certas modalidades preferidas de acordo com a invenção, existem polipeptídeos de fusão de Mycobacterium tuberculosis fornecidos, e polinucleo- tídeos codificando polipeptídeos de fusão. O polipeptídeo de fusão e as proteínas de fusão referem-se a um polipeptídeo que tem pelo menos dois polipeptídeos heteró- logos do Mycobacterium sp., tais como polipeptídeos de Mycobacterium tuberculosis, covalentemente ligados, diretamente ou via um ligante de aminoácido. Os polipeptídeos que formam a proteína de fusão são tipicamente ligados da terminação C para a terminação N, embora eles também possam ser ligados da terminação C para a terminação C, da terminação N para a terminação N, ou da terminação N para a terminação C. Os polipeptídeos da proteína de fusão podem estar em qualquer ordem. Os polipeptídeos de fusão ou as proteínas de fusão também podem incluir variantes modificadas de modo conservador, variantes polimorfas, alelos, mutantes, subsequências, homólogo de interespécies, e fragmentos imunogênicos dos antígenos que compõem a proteína de fusão. Os antígenos de Mycobacterium tuberculosis são descritos em Cole et al. Nature 393:537 (1998), que descrevem o genoma inteiro do Mycobacterium tuberculosis. Os antígenos de outras espécies de Mycobacterium que equivalem a antígenos de Mycobacterium tuberculosis podem ser identificados, por exemplo, usando algoritmos de comparação de sequência, tal como aqui descrito, ou outros métodos conhecidos daqueles com habilidade na técnica, por exemplo, ensaios de hibridização e ensaios de ligação de anticorpo.
[00313]Os polipeptídeos de fusão de acordo com a invenção geralmente compreendem pelo menos dois polipeptídeos antigênicos tal como aqui descrito, e podem compreender também outras sequências não relacionadas, tais como uma sequência que auxilia no fornecimento de epítopos T auxiliares (um parceiro de fusão imunológico), preferivelmente epítopos T auxiliares reconhecidos por humanos, ou que auxiliam na expressão da proteína (um melhorador de expressão) em rendimentos mais altos do que a proteína recombinante nativa. Alguns parceiros de fusão preferidos são tanto melhoradores imunológicos e parceiros de fusão melhoradores de expressão. Outros parceiros de fusão podem ser selecionados para aumentar a solubilidade da proteína ou permitir à proteína a ser visada nos compartimentos intracelulares desejados. Ainda adicionalmente os parceiros de fusão incluem marcações de afinidade, que facilitam a purificação da proteína.
[00314]As proteínas de fusão podem ser geralmente preparadas usando técnicas padrão. Preferivelmente, uma proteína de fusão é expressada como uma proteína recombinante. Por exemplo, as sequências de DNA codificando os compo-nentes de polipeptídeo de uma fusão desejada que podem ser montadas separada-mente, e ligar em um vetor de expressão apropriado. A extremidade 3' da sequência de DNA que codifica um componente polipeptídeo é ligada, com ou sem um peptídio ligante, à extremidade 5' de uma sequência de DNA que codifica o segundo compo-nente do polipeptídeo para que as estruturas de leitura das sequências estejam em fase. Isto permite a tradução para uma proteína de fusão única que conserva a atividade biológica de polipeptídeos de componente de ambos.
[00315]Uma sequência de ligante de peptídio pode ser empregada para separar os primeiros e segundos componentes de polipeptídeo por uma distância suficiente para assegurar que cada polipeptídeo se dobra nas suas estruturas secundárias e terciárias, se desejado.
[00316]Tal sequência de ligante de peptídio é incorporada na proteína de fusão usando técnicas padrão bem conhecidas na técnica. Certas sequências de ligante de peptídio podem ser escolhidas baseadas nos seguintes fatores: (1) a sua capacidade de adotar uma conformação extensa flexível; (2) a sua incapacidade de adotar uma estrutura secundária que possa interagir com epítopos funcionais nos primeiros e segundos polipeptídeos; e (3) a falta de resíduos hidrofóbicos ou carregados que poderiam reagir com o polipeptídeo epítopos funcionais. As sequências de ligante de peptídio preferidas contêm Gly, Asn e resíduos Ser. Outro perto de aminoácidos neutros, tais como Thr e Ala também pode ser usado na sequência de ligante. As sequências de aminoácido que podem ser utilmente empregadas como ligantes incluem os divulgados em Maratea et al., Gene 40:39 46 (1985); Murphy et al., Proc. Natl. Acad. ScL EUA 83:8258 8262 (1986); Patente dos Estados Unidos N° US. 4.935.233 e Patente dos Estados Unidos N° US. 4.751.180. A sequência de ligante pode ser geralmente de 1 a aproximadamente 50 aminoácidos no comprimento. As sequências de ligante não são necessárias quando os primeiros e segundos polipeptídeos têm regiões que não são de aminoácido de terminação N essenciais que podem ser usadas para separar os domínios funcionais e prevenir a interferência esférica.
[00317]As sequências de DNA ligar são operavelmente ligado a elementos reguladores transcricionais ou de translação adequados. Os elementos reguladores responsáveis pela expressão do DNA são localizados somente 5' à sequência de DNA que codifica os primeiros polipeptídeos. Semelhantemente pare codons necessitado terminar a tradução e o sinal de terminação de transcrição é 3 somente presentes' à sequência de DNA que codifica o segundo polipeptídeo.
[00318]Dentro de modalidades preferidas, um parceiro de fusão imunológico para uso em um polipeptídeo de fusão de acordo com a invenção é derivado da proteína D, uma proteína superficial da bactéria negativa pelo grama influenza de Haemophilus B (WO 91/18926). Preferivelmente, uma proteína D derivado compreende aproximadamente o primeiro terço da proteína (por exemplo, o primeira terminação N 100110 aminoácidos), e uma proteína D derivado pode ser lipidated. Dentro de certas modalidades preferidas, 109 primeiros resíduos de uma lipoproteína D parceiro de fusão estão incluídos na terminação N para prover o polipeptídeo de exogenous adicional T epítopos de célula e aumentar o nível de expressão em E. coli (assim funcionando como um melhorador de expressão). A cauda de lipídio assegura a ótima apresentação do antígeno a células de presença de antígeno. Outros parceiros de fusão incluem a proteína não estrutural do vírus influenzae, NS1 (hemagluti- nin). Tipicamente, a terminação N 81 aminoácidos são usados, embora os fragmentos diferentes que incluem epítopos de T-ajudante possam ser usados.
[00319]Em outra modalidade, um parceiro de fusão imunológico compreende uma sequência de aminoácidos derivou da proteína conhecida como LYTA, ou porção do mesmo (preferivelmente um terminação C porção). LYTA é derivado do Es- treptococo pneumoniae, que sintetiza um N-acetil-L-alanina amidase conhecido como amidase LYTA (codificado pelo gene LytA; Gene 43:265-292 (1986)). LYTA é uma autolisina que especificamente degrada certas ligações na cadeia principal de peptidoglicano. Terminação C o domínio da proteína LYTA é responsável pela afinidade à colina ou a análogos de colina alguns, tais como DEAE. Esta propriedade foi explorada para o desenvolvimento de E. colí C-LYTA plasmídios expressam úteis para expressão das proteínas de fusão. A purificação de proteínas híbridas contendo o fragmento C-LYTA na estação final de amina foi descrita (ver a Biotecnologia '70:795-798 (1992)). Dentro de modalidade preferida, uma porção repetida de LYTA pode ser incorporada em uma proteína de fusão. Uma porção repetida é encontrada na região da terminação C que inicia no resíduo 178. Uma porção repetida particularmente preferida incorpora os resíduos de 188 a 305.
[00320]Em geral, os polipeptídeos e os polipeptídeos de fusão (bem como os seus polinucleotídeos de codificação) são isolados. Um polipeptídeo ou polinucleotídeo "isolado" são aquele que é removido dhis ambiente original. Por exemplo, uma proteína que ocorre naturalmente é isolada se ela for separada de pouco ou todos dos materiais coexistem no sistema natural. Preferivelmente, tais polipeptídeos são pelo menos aproximadamente 90 % puros, mais preferivelmente pelo menos aproximadamente 95 % puros e bem mais preferivelmente pelo menos aproximadamente 99 % puros. Considera-se que um polinucleotídeo é isolado se, por exemplo, for clonado em um vetor que não é uma parte do ambiente natural.
B. Composições de Polinucleotídeo.
[00321]A presente invenção também fornece polinucleotídeos isolados, par-ticularmente aqueles codificando os polipeptídeos de fusão de acordo com a invenção, bem como composições compreendendo tais polinucleotídeos. Tal como aqui utilizado, os termos "DNA" e "polinucleotídeo" e "ácido nucléico" referem-se a uma molécula de DNA que foi isolada livre do DNA genômico total de uma espécie em particular. Desse modo, um segmento de DNA que codifica um polipeptídeo refere- se a um segmento de DNA contendo uma ou mais sequências de codificação ainda é substancialmente isolado longe de, ou purificado livre de, total genomic o DNA das espécies das quais o segmento de DNA é obtido. Incluído dentro dos termos "segmento de DNA" e "polinucleotídeo" são segmentos de DNA e fragmentos de menor de tais segmentos, e também vetores recombinantes, incluindo, por exemplo, plas- mídios, cosmídios, fagomídios, fago, vírus, e assim por diante.
[00322]Como será entendido pelos versados na técnica, as sequências de polinucleotídeo da presente invenção podem incluir sequências genômicas, as se-quências extra-genômicas e codificadas pelo plasmídio e o menor projetaram seg-mentos genéticos que expressam, ou podem ser adaptados para expressar, proteínas, polipeptídeos, peptídios e assim por diante. Tais segmentos podem ser naturalmente isolados, ou modificados sinteticamente pela mão do homem.
[00323]Como será reconhecido pelo técnico versado, os polinucleotídeos podem ser de fita simples (codificação ou antisentido) ou de fita dupla, e podem ser DNA (genômico, cDNA ou sintético) ou moléculas de RNA. Sequências de codificação adicional ou de não codificação, mas não precisam, podem estar presentes dentro de um polinucleotídeo da presente invenção, e um polinucleotídeo, mas não precisam, pode ser ligado a outras moléculas e/ou materiais de suporte.
[00324]Os polinucleotídeos podem compreender uma sequência nativa (isto é, uma sequência endógena que codifica um antígeno de Mycobacterium ou uma porção do mesmo) ou pode compreender uma variante, ou equivalente funcional biológico ou antigênico de tal sequência. As variantes de polinucleotídeo podem conter uma ou mais substituições, adições, eliminações inserções de and/or, como também descrito abaixo, preferivelmente de tal modo que a imunogenicidade do polipeptídeo codificado seja não diminuído, em relação à proteína nativa. O efeito sobre a imunogenicidade do polipeptídeo codificado pode ser geralmente avaliado tal como aqui descrito. O termo "variantes" também abrange genes homólogos da origem xe- nogênica.
[00325]Em modalidades adicionais, a presente invenção fornece polinucleo-tídeos isolados os comprimentos compreendendo diferentes dos esticamentos contí-guos da sequência idêntica a ou complementar a uma ou mais das sequências aqui divulgadas. Por exemplo, os polinucleotídeos são fornecidos pela presente invenção que compreendem pelo menos aproximadamente 15, 20, 30, 40, 50, 75, 100, 150, 200, 300, 400, 500 ou 1000 ou mais de um ou mais de nucleotídios contíguo das sequências aqui divulgadas bem como todos os comprimentos intermediários lá entre. Compreender-se-á prontamente que "comprimentos intermediários", neste contexto, significam quaisquer comprimentos entre os valores cotados, tal como 16, 17, 18, 19, etc.; 21, 22, 23, etc.; 30, 31, 32, etc.; 50, 51, 52, 53, etc.; 100, 101, 102, 103, etc.; 150, 151, 152, 153, etc.; incluindo todos os números inteiros até 200 500; 500 1.000, e assim por diante.
[00326]Os polinucleotídeos da presente invenção, ou fragmentos dos mesmos, independente do comprimento da própria sequência de codificação, podem ser combinados com outras sequências de DNA, tais como promotores, sinais de polia- denilação, sítios de enzima de restrição adicionais, múltiplos sítios de clonagem, outros segmentos de codificação, e assim por diante, de tal modo que his comprimento total pode variar consideravelmente. É desse modo contemplado que pode ser empregado um fragmento de polinucleotídeo de quase qualquer comprimento, com o comprimento total sendo preferivelmente limitado pela facilidade de preparação e uso no protocolo do DNA recombinante desejado.
[00327]Além disso, será apreciado por aqueles com habilidade normal na técnica que, em consequência da degeneração do código genético, existem muitas sequências de nucleotídio codificando um polipeptídeo tal como aqui descrito. Alguns desses polinucleotídeos carregam a homologia mínima com a sequência de nucleotídio de qualquer gene nativo. Todavia, os polinucleotídeos que variam devido a diferenças no uso do codon são especificamente contemplados pela presente invenção, por exemplo, polinucleotídeos que são otimizados para seleção de códon humano e/ou de primata. Além disso, alelos dos genes compreendendo o fornecimento de sequências de polinucleotídeo aqui são dentro do alcance da presente invenção.
[00328]Alelos são genes endógenos que são alterados como resultados de uma ou mais mutações, tais como eliminações, adições e/ou substituições de nucle- otídios. O mRNA resultante e a proteína podem, mas não precisam, ter uma estrutura ou função alterada. Os alelos podem ser identificados usando técnicas padrão (tais como hibridização, amplificação e/ou comparação da sequência de banco de dados).
[00329]Os polinucleotídeos de Mycobacterium e as fusões dos mesmos podem ser preparados, manipulados e/ou expressados utilizando qualquer uma das várias técnicas bem estabelecidas conhecidas e disponíveis na técnica.
[00330]Por exemplo, as sequências de polinucleotídeo ou os fragmentos dos mesmos codificando polipeptídeos, ou proteínas de fusão ou equivalentes funcionais do mesmo, de acordo com a invenção podem ser usados em moléculas de DNA re-combinantes para dirigir a expressão de um polipeptídeo em células hospedeiras apropriadas. Devido à degeneração inerente do código genético, outras sequências de DNA codificando substancialmente a mesma, ou uma sequência de aminoácidos funcionalmente equivalente, podem ser produzidas e estas sequências podem ser usadas para clonar e expressar um polipeptídeo fornecido.
[00331]Como será entendido por aqueles com habilidade na técnica, pode ser vantajoso em exemplos alguns produzir sequências de nucleotídio codificam o polipeptídeo que possuem não naturalmente ocorrendo codons. Por exemplo, codons preferido por um hospedeiro procariótico ou eucariótico particular pode ser selecionado para aumentar a taxa da expressão da proteína ou produzir uma cópia de RNA recombinante que tenha propriedades desejáveis, tais como uma meia-vida que é mais longa do que aquela de uma cópia gerada da sequência que ocorre naturalmente.
[00332]Além disso, as sequências de polinucleotídeo da presente invenção podem ser projetadas usando métodos geralmente conhecidos na técnica para alterar sequências de codificação de polipeptídeo para um a variedade de razões, incluindo mas não limitadas a, alterações que modificam a clonagem, processamento, expressão a imunogenicidade de e/ou do produto genético.
[00333]Para expressar um polipeptídeo desejado, uma sequência de nucleotídio que codifica o polipeptídeo, ou equivalente funcional, pode ser inserida no vetor de expressão apropriado, isto é, um vetor contendo os elementos necessários da transcrição e a tradução da sequência de codificação inserida. Os métodos que são bem conhecidos aos versados na técnica podem ser usados para construir vetores de expressão contendo sequências codificando um polipeptídeo de interesse e apropriar-se de elementos de controle transcricionais e de translação. Estes métodos incluem técnicas de DNA in vitro recombinantes, técnicas sintéticas, e recombi- nação in vivo genética. Tais técnicas são descritas em Sambrook et al., Molecular Cloning, A Laboratory Manual (1989), e Ausubel et al., Current Protocols in Molecular Biology (1989).
[00334]Vários sistemas de vetor/hospedeiro de expressão são conhecidos e podem ser utilizados para conter e expressar sequências de polinucleotídeo. Estes incluem, mas são não limitados a, microrganismos, tais como bactérias transformadas com bacteriophage recombinante, plasmídio, ou vetores de expressão de cos- mídio de DNA; a levedura transformada com vetores de expressão da levedura; os sistemas de célula de inseto infectam com vetores de expressão de vírus (por exemplo, baculovirus); sistemas de célula de planta transformados com vetores de expressão de vírus (por exemplo, vírus de mosaico de couve-flor, CaMV; vírus de mosaico do tabaco, TMV) ou com vetores de expressão bacterianos (por exemplo, Ti ou plasmídios pBR322); ou sistemas de célula dos animais.
[00335]"Os elementos de controle" ou "sequências reguladoras" presentes em um vetor de expressão são aquelas regiões não traduzidas dos melhoradores do vetor, promotores, regiões não traduzidas 5' e 3', que interagem com as proteínas celulares do hospedeiro para executar a transcrição e a tradução. Tais elementos podem variar na sua força e especificidade. Dependendo do sistema de vetor e hospedeiro utilizado, uma quantidade qualquer de transcrições adequadas e de elementos de tradução, incluindo promotores constitutivos e induzívéis, pode ser usada. Por exemplo, quando se clonam sistemas bacterianos, promotores induzívéis, tais como o promotor híbrido lacZ do PBLUESCRIPT fagomídio (Stratagene, La Jolla, Califórnia.) ou plasmídio PSPORT1 (Gibco BRL, Gaithersburg, Md.) e assim por diante, podem ser usados. Em sistemas de célula de mamífero, os promotores de genes mamíferos ou de vírus mamíferos são geralmente preferidos. Se for necessário gerar uma linhagem celular contendo múltiplas cópias da sequência que codifica um polipeptídeo, os vetores baseados em SV40 ou EBV podem ser vantajosamente usados com um marcador selecionável apropriado.
[00336]Em sistemas bacterianos, diversos vetores de expressão podem ser selecionados, dependendo do uso destinado para o polipeptídeo expressado. Por exemplo, quando grandes quantidades são necessárias, vetores que expressão de alto nível direta de proteínas de fusão que são prontamente purificadas podem ser usados. Tais vetores incluem, mas são não limitados a, clonagem multifuncional de E. coli e vetores de expressão, tais como BLUESCRIPT (Stratagene), no qual a sequência que codifica o polipeptídeo de interesse pode ser ligada no vetor na estrutura com as sequências da MET da terminação amina e 7 resíduos subsequentes de D-galactosidase para que uma proteína híbrida seja produzida; vetores pi N (Van Heeke & Schuster, J. Biol. Chem. 264:5503 5509 (1989)); e assim por diante. Vetores pGEX (Promuito, Madison, Wis.) também pode ser usado para expressar polipeptídeos estranhos como proteínas de fusão com a Glutationa S-transferase (GST). Em geral, tais proteínas de fusão são solúveis e podem ser facilmente purificadas de células lisadas pela adsorção em pérolas de agarose de glutationa seguidos pela eluição na presença de glutationa livre. As proteínas feitas em tais sistemas podem ser projetadas para incluir heparina, trombina, ou os sítios de divagem da protease do Fator XA para que o polipeptídeo clonado de interesse possa ser liberado da porção GST à vontade. Na levedura, o Saccharomyces cerevisiae, diversos vetores contendo promotores constitutivos ou induzívéis, tais como fator alfa, álcool oxidase, e PGH podem ser usados. Para revisões, ver Ausubel et al. (supra) e Grant et al., Methods Enzymol 153:516-544 (1987).
[00337]Em casos onde os vetores de expressão de planta são usados, a ex-pressão de sequências codificando polipeptídeos pode ser dirigida por qualquer uma das diversas promotores. Por exemplo, os promotores virais, tais como os 35 e os promotores de 19 do CaMV podem ser usados sozinhos ou em combinação com a sequência líder de ômega de TMV (Takamatsu, EMBO J. 6:307-311 (1987)). Alterna- tivamente, os promotores de plantas, tais como a pequena subunidade de RUBISCO ou promotores de choque de calor podem ser usados (Coruzzi et al., EMBO J. 3:1671-1680 (1984); Broglie et al., Science 224:838-843 (1984); e Winter et al., Results Probl. Cell Differ. 17:85-105 (1991)). Estes construtores podem ser introduzidos nas células das plantas por transformação direta de DNA ou transfecção mediada no agente patogênico. Tais técnicas são descritas em algumas geralmente diferentes revisões disponíveis (ver, por exemplo, Hobbs em McGraw Hill, Yearbook of Science and Technology, páginas 191-196 (1992)). Um sistema de inseto também pode ser usado para expressar um polipeptídeo de interesse. Por exemplo, em um tal sistema, o vírus poliedrose nuclear Autographa californica (AcNPV) é usado como um vetor para expressar genes estranhos nas células da Spodoptera frugiperda ou em larvas da Trichoplusia. As sequências codificando o polipeptídeo podem ser clona- das em uma região não essencial do vírus, tal como o gene poliedrina, e colocadas sob controle do promotor da poliedrina. A inserção bem sucedida da sequência que codifica o polipeptídeo produzirá o gene poliedrina inativo e produzirá o vírus recombinante que necessita de proteína de revestimento. Os vírus recombinantes então podem ser usados para infeccionar, por exemplo, células de S. frugiperda ou larvas Trichoplusia nas quais o polipeptídeo de interesse pode ser expressado (Engelhard et al., Proc. Natl. Acad. Sei. U.S.A. 97:3224-3227 (1994)).
[00338]Em células hospedeiras de mamíferos, diversos sistemas de expressão baseados em virus estão geralmente disponíveis. Por exemplo, em casos onde um adenovirus é usado como um vetor de expressão, as sequências codificando um polipeptídeo de interesse podem ser ligadas em um complexo de transcri- ção/tradução de adenovirus consistindo do promotor tardio e da sequência líder tripartite. A inserção em um E1 não essencial ou a região E3 do genoma virai pode ser usada para obter um vírus viável que é capaz de expressar o polipeptídeo em células de hospedeiros infectados (Logan & Shenk, Proc. Natl. Acad. Sci. U.S.A. 87:3655-3659 (1984)). Além disso, os melhoradores de transcrição, tais como o me- Ihorador do vírus de sarcoma Rous (RSV), podem ser usados para aumentar a expressão em células de hospedeiros mamíferos.
[00339]O sinal de iniciação específico também pode ser usado para atingir a tradução mais eficiente de sequências codificando um polipeptídeo de interesse. Tal sinal inclui o codon de iniciação ATG e sequências adjacentes. Em casos onde se-quências codificando o polipeptídeo, a sua iniciação codon, e à montante as se-quências são inseridas no vetor de expressão apropriado, nenhum sinal de controle transcricional ou de translação adicional pode ser necessário. Entretanto, em casos onde somente codificar sequência, ou porção do mesmo, é inserido, exogenous sinal de controle de translação que inclui a iniciação ATG o codon deve ser fornecido. Além disso, a iniciação codon deve estar na estrutura de leitura correta para assegurar a tradução da inserção inteira. Elementos exógenos de translação e iniciação codons podem ser de origens diversas, tanto naturais e sintéticas. A eficiência da expressão pode ser melhorada pela inclusão de melhoradores que são apropriados para o sistema de célula particular que é usado, tais como os descritos na literatura (Scharf, et al., Results Probl. Cell Differ. 20:125-162 (1994)).
[00340]Além disso, uma cepa de célula hospedeira pode ser escolhida pela sua capacidade de modular a expressão das sequências inseridas ou processar a proteína expressada da maneira desejada. Tais modificações do polipeptídeo incluem, mas são não limitadas a, acetilação, carboxilação, glicação, fosforilação, lipida- ção, e acilação. O processamento pós-translação que cliva uma “prepro" forma da proteína também pode ser usada para facilitar a inserção correta, e/ou a função de dobra. As células de hospedeiros diferentes, tais como CHO, HeLa, MDCK, HEK293, e W138, possuindo maquinismo celular específico e mecanismos característicos de tais atividades pós-translação, podem ser escolhidos para assegurar a modificação correta e o processamento da proteína estranha.
[00341]Para a produção de longo prazo, de alta rentabilidade de proteínas recombinantes, é geralmente preferida a expressão estável. Por exemplo, a linhagem de células que de maneira estável expressam um polinucleotídeo de interesse podem ser transformadas usando vetores de expressão que podem conter réplicas de origens virais e/ou elementos de expressão endógenos e um gene de marcador selecionável no mesmo ou sobre uma separar o vetor. Depois da introdução do vetor, pode permitir-se que células cresçam durante 1 a 2 dias em um meio enriqueci- do antes que eles sejam trocados ao meio seletivo. O objetivo do marcador selecio- nável é conferir a resistência à seleção, e a sua presença permite o crescimento e a recuperação de células que com sucesso expressam as sequências introduzidas. Os clones resistentes de células transformadas de maneira estável podem ser proliferados usando técnicas de cultura de tecido apropriadas para o tipo de célula.
[00342]O número qualquer de sistemas de seleção pode ser usado para re-cuperar a linhagem de células transformado. Estes incluem, mas são não limitados a, o herpes simplex quinase de timidina de vírus (Wigler et al., Cell 77:223-232 (1977)) e adenina fosforribosil transferase (Lowy et al., Ce//22:817-823 (1990)) genes que podem ser empregados em células tk- ou aprt-, respectivamente. Também, o antimetabólito, o antibiótico ou a resistência de herbicida podem ser usados como a base da seleção; por exemplo, dhfr que confere a resistência a metotrexato (Wigler et al., Proc. Natl. Acad. ScL U.S.A. 77:3567-70 (1980)); o npt, que confere a resistência ao aminoglicosídios, neomicina e G-418 (Colbere-Garapin et al., J. Mol. Biol. 750:1-14 (1981)); e o ais, ou apropriado, que conferem a resistência ao clorosulfuron e fosfinotricina acetiltransferase, respectivamente (Murry, supra). Genes adicionais selecionáveis foram descritos, por exemplo, trpB, que permite a células utilizar o in- dol no lugar do triptofano, ou o hisD, que permite a células utilizar histinol no lugar da histidina (Hartman & Mulligan, Proc. Natl. Acad. ScL U.S.A. 85:8047 - 51 (1988)). O uso de marcadores visíveis ganhou a popularidade com marcadores tais como anto- cianinas, β-glucuronidase e seu substrato GUS, e luciferase e seu substrato de luci- ferina, sendo amplamente usado não somente para identificar transformantes, mas também quantificar a quantidade da expressão da proteína transiente ou estável atribuível a um sistema de vetor específico (Rhodes et al., Methods Mol. Biol. 55:121-131 (1995)).
[00343]Vários protocolos para detectar e medir a expressão de produtos co-dificados pelo polinucleotídeo, usando anticorpos policlonais ou monoclonais especí- ficos para o produto são conhecidos na técnica. Os exemplos incluem o ensaio imu- nosorbent ligado à enzima (ELISA), radioimunoensaio (RIA), e a classificação de célula ativada por fluorescência (FACS). Estes e outros ensaios são descritos, entre outros lugares, em Hampton et al., Sorológico Methods, a Laboratory Manual (1990) e Maddox et al., J. Exp. Med. 758:1211-1216 (1983). Uma variedade ampla de marcador e técnicas de conjugação é conhecida pelos versados na técnica e pode ser usada em vários ensaios de aminoácido e ácido nucléico. O meio para produzir o marcador de hibridização ou as sondas de PCR para detectar sequências relacionadas a polinucleotídeos inclui a oligomarcação, tradução de fresta, marcador da extremidade ou amplificação PCR usando um nucleotídio marcado. Alternativamente, as sequências, ou quaisquer porções do mesmo podem ser clonadas em um vetor para produção de uma sonda de mRNA. Tais vetores são conhecidos na técnica, estão comercialmente disponíveis, e podem ser usados para sintetizar sondas de RNA in vitro pela adição de uma polimerase apropriada de RNA, tais como T7, T3, ou SP6 e marcador de nucleotídios. Estes procedimentos podem ser conduzidos usando vários kits comercialmente disponíveis. As moléculas de repórter adequadas ou o marcador, que pode ser usado incluem radionuclídios, enzimas, fluorescentes, quimioluminescência, ou agentes cromogênicos bem como substratos, cofactores, inibidores, partículas magnéticas, e assim por diante.
[00344]As células de hospedeiros transformados com uma sequência de po-linucleotídeo de interesse podem ser cultivadas sob condições adequadas para a expressão e a recuperação da proteína da cultura de célula. A proteína produzida por uma célula recombinante pode ser secretada ou contida intracelularmente dependendo da sequência e/ou do vetor usado. Como será entendido por aqueles com habilidade na técnica, os vetores de expressão contendo os polinucleotídeos de acordo com a invenção podem ser projetados para conter sequências de sinal que a secreção direta do polipeptídeo codificado por uma membrana de célula procariótica ou eucariótica. Outras construções recombinantes podem ser usadas para unir sequências codificando um polipeptídeo de interesse à sequência de nucleotídio que codifica um domínio de polipeptídeo que facilitará a purificação de proteínas solúveis.
[00345]Além de métodos de produção recombinantes, os polipeptídeos de acordo com a invenção, e os fragmentos dos mesmos, podem ser produzidos pela síntese de peptídio direta usando técnicas de fase sólida (Merrifield, J. Biol. Chem. Soc. 85:2149 - 2154 (1963)). A síntese de proteína pode ser realizada usando técnicas manuais ou pela automação. A síntese automatizada pode ser atingida, por exemplo, usando Biosystems Applyed 431 um Sintetizador de Peptídio (Perkin Elmer). Alternativamente, os fragmentos diferentes podem ser quimicamente sintetizados separadamente e combinaram métodos químicos usados para produzir a molécula de comprimento total.
C. Produto Farmacêutico e Composições de Vacina.
[00346]Em outro aspecto, a presente invenção de um ou mais de formulações de preocupações do polinucleotídeo, polipeptídeo ou outras composições aqui divulgadas em soluções farmaceuticamente aceitáveis ou fisiologicamente aceitáveis para administração a uma célula ou um animal, sozinho, ou em combinação com uma ou mais outras modalidades de terapia. Tais composições farmacêuticas são particularmente preferidas para uso como vacinas quando formuladas com um imu- noestimulante adequado / sistema adjuvante. As composições são também adequadas para uso em um contexto diagnóstico.
[00347]Também se entenderá que, se desejado, as composições de acordo com a invenção podem ser administradas em combinação com outros agentes também, tal como, por exemplo, outras proteínas ou polipeptídeos ou agentes ativos do farmaceuticamente diferentes. Não exista praticamente nenhum limite de outros componentes que também pode ser incluíram, contanto que os agentes adicionais não causem um efeito averso significante sobre os objetivos de acordo com a invenção.
[00348]Em certas modalidades preferidas as composições de acordo com a invenção são usadas como vacinas e são formuladas em combinação com um ou mais imunoestimulantes. Um imunoestimulante pode ser substância qualquer que melhora ou potencializa uma resposta imune (anticorpo e/ou mediado na célula) a um antígeno exogenous. Os exemplos de imunoestimulantes incluem adjuvantes, microesferas biodegradáveis (por exemplo, galactide poliláctico) e lipossomas (no qual o composto é incorporado; ver, por exemplo, Fullerton, Patente dos Estados Unidos N° US. 4.235.877). A preparação de vacina é geralmente descrita em, por exemplo, Powell & Newman, editores, Vaccine Design (a subunidade e aproximação de adjuvante) (1995).
[00349]Qualquer de vários imunoestimulantes pode ser empregado nas vacinas do a presente invenção. Por exemplo, um adjuvante pode estar incluído. Muitos adjuvantes contêm uma substância projetada para proteger o antígeno de catabolism rápido, tal como hidróxido de alumínio ou óleo mineral, e um estimulador de respostas imunes, tais como lipídio A (natural ou sintético), espécies proteins derivadas de Mycobacterium bortadella ou Mycobacterium pertussis. Os adjuvantes adequados são comercialmente disponíveis como, por exemplo, o Adjuvante Incompleto de Freund e Concluem o Adjuvante (Laboratórios de Difco, a Detroit, Mich.); Adjuvante de Merck 65 (Merck and Company, Inc, Rahway, N.J.); AS-2 e derivados do mesmo (SmithKline Beecham, Filadélfia, Papai) ; CWS, TDM, Leif, sais de alumínio, tais como gel de hidróxido de alumínio (alume) ou fosfato de alumínio; sais de cálcio, ferro ou zinco; uma suspensão insolúvel de tirosina acylated; açúcar de acylated; cationically ou polissacarídios de derivada anionically; poliphosphazenes; microesferas biodegradáveis; lipídio A de monofosforil e A. Cytokines quil, tal como GM-CSF ou interleucina 2, -7, ou -12, também pode ser usado como adjuvantes.
[00350]Em certas modalidades preferidas, o adjuvante usado na a presente invenção é um adjuvante lipídio A glicopiranosil (GLA) adjuvante, como descrito no Pedido Patente dos Estados Unidos pendente Serial N° 11/862.122, a divulgação de que é incorporado aqui pela referência em sua totalidade. Por exemplo, certos com-postos GLA de interesse são representados pela seguinte fórmula:
onde: R1, R3, R5 e R6 são alquil(Ci 1-C20); e R2 e R4 são alquil(Ci2-C20). Em uma modalidade mais particular, R1, R2, R3, R4, R5 e R6 são Cu. Outros adjuvantes ilustrativos úteis no contexto de acordo com a invenção incluem agonistas dos Toll- like receptor, tais como agonistas de TLR7, agonistas de TLR7/8, e assim por diante. Em todo o caso outros adjuvantes ilustrativos incluem imiquimod (IMQ), gardiquimod (GDQ), resiquimod (RSQ), e compostos relacionados.
[00351]Certain preferiu que as vacinas empreguem sistemas de adjuvante projetados para induzir uma resposta imune predominantemente do tipo de Th1. Os altos níveis de citoquinas de tipo de th1-(por exemplo, IFN-y, TNF, IL-2 e IL-12) tendem a favorecer a indução de respostas imunes mediadas de célula a um antígeno administrado. Ao contrário os altos níveis de citoquinas do tipo Th2 (por exemplo, IL- 4, IL-5, IL-6 e IL-10) tendem a favorecer a indução de respostas imunes humorais. O seguinte pedido de uma vacina contanto que aqui, um paciente suportará uma resposta imune que inclui respostas do tipo Th2 e Th1. Dentro de modalidade preferida, na qual uma resposta é predominantemente Th1 -tipo, o nível de citoquinas do tipo Th1 aumentará até um maior ponto do que o nível de citoquinas do tipo Th2. Os níveis destas citoquinas podem ser prontamente avaliados usando ensaios padrão. Para um a revisão das famílias de citoquinas, ver Mossman & Coffman, Ann. Rev. Immunol. 7:145-173 (1989).
[00352]Certos adjuvantes para uso na obtenção predominantemente resposta do tipo Th1 incluem, por exemplo, uma combinação do lipídio A monofosforil, pre-ferivelmente 3-de-O-acylated monofosforil lipídio A (3D-MPL®), em conjunto com um sal de alumínio (Patente dos Estados Unidos N° US. 4.436.727; 4.877.611; 4.866.034; e 4.912.094). Oligonucleotídios contendo CpG (no qual o CpG dinucleotí- dio é não metilado) também induz um predominantemente resposta de Th1. Tais oligonucleotídios são bem conhecidos e são descritos, por exemplo, na WO 96/02555, WO 99/33488 e Patente dos Estados Unidos N° US. 6.008.200 e 5.856.462. as sequências de DNA de Immunostimulatory são também descritas, por exemplo, por Sato et al., Science 273:352 (1996). Outro adjuvante ilustrativo compreende uma saponina, tal como Quil A, ou derivados do mesmo, incluindo QS21 e QS7 (Aquila Biopharmaceuticals Inc, Framingham, massa.) ; Escin; digitonina; ou Gypsophila ou Chenopodium quinoa saponinas. Outras formulações ilustrativas incluem mais do que uma saponina nas combinações de adjuvante da presente invenção, por exemplo as combinações dos pelo menos dois dos seguintes grupos compreendendo QS21, QS7, Quil A, escina, ou digitonina. Em uma modalidade particular, o sistema de adjuvante inclui a combinação de um lipídio A monofosforil e um derivado de saponina, tal como a combinação de adjuvante QS21 e 3D-MPL®, como descrito na WO 94/00153, ou uma composição menos reatogênica onde o QS21 é extinto com o colesterol, tal como descrito na WO 96/33739. Outras formulações compreendem uma emulsão de óleo na água e tocoferol. Outra formulação de adjuvante que emprega QS21, 3D-MPL® adjuvante e tocoferol em uma emulsão de óleo na água é descrita na WO 95/17210.
[00353]outro sistema de adjuvante melhorado envolve a combinação de um oligonucleotídio contendo CpG e um derivado de saponina como divulgado na WO 00/09159. Outros adjuvantes ilustrativos incluem Montanide ISA 720 (Seppic, França), SAF (Novartis, Califórnia., Estados Unidos), ISCOMS (CSL), MF-59 (Chiron), a série SBAS de adjuvantes (por exemplo, SBAS-2, AS21, AS2”, SBAS-4, ou SBAS6, disponível pela GlaxoSmithKline, Rixensart, Bélgica), Desintoxicação, RC-529 (GlaxoSmithKline, Hamilton, Mont.) e outro aminoalkyl glucosaminide 4 fosfatos (AGPs), tais como os descritos nos Estados Unidos pendentes Pedido de Patente Ser. Os números 08/853,826 e 09/074,720, as divulgações do qual são incorporadas aqui por referência nas suas totalidades, e adjuvantes de éter de polioxietileno, tais como os descritos na WO 99/52549A1.
[00354]As composições de acordo com a invenção podem também, ou al-ternativamente, compreender células T específicas para um antígeno de Mycobacte-rium. Tais células podem ser geralmente preparadas in vitro ou ex vivo, usando pro-cedimentos padrão. Por exemplo, células T pode ser isolado de medula de osso, sangue periférico, ou uma fração da medula de osso ou sangue periférico de um paciente. Alternativamente, as células T podem ser derivado de seres humanos relacionados ou não relacionados, mamíferos não humanos, linhagem de células ou culturas. As células T podem ser estimuladas com um polipeptídeo de acordo com a invenção, polinucleotídeo que codifica tal polipeptídeo, e/ou uma célula de presença de antígeno (APC) que expressa tal polipeptídeo. Tal estimulação é realizada sob condições e durante algum tempo suficiente para permitir a geração de células T que são específicas para o polipeptídeo. Preferivelmente, o polipeptídeo ou o polinucleotídeo estáêm presente dentro de um veículo de liberação, tal como uma microesfera, para facilitar a geração de células T específicas.
[00355]As células T são consideradas como sendo específicas para um o polipeptídeo de acordo com a invenção se as células T especificamente proliferarem, citoquinas de segregar ou matarem células de objetivo cobertas do polipeptídeo ou expressão de um gene que codifica o polipeptídeo. A especificidade de célula T pode ser avaliada usando qualquer de várias técnicas padrão. Por exemplo, dentro de um ensaio de liberação de cromo ou ensaio de proliferação, um índice de estimulação de mais do que dois aumento de prega no lise a proliferação de and/or, em comparação com o controle negativo, indica a especificidade de célula T. Tais ensaios podem ser realizadas, por exemplo, como descrito em Chen et al., Cancer Res. 54:1065-1070 (1994)). Alternativamente, a detecção da proliferação de células T pode ser realizada por várias técnicas conhecidas. Por exemplo, T proliferação de célula pode ser detectado medindo uma taxa aumentada da síntese de DNA (por exemplo, por culturas marcador do pulso de células T com a timidina tritiated e medir a quantidade da timidina tritiated incorporou-se no DNA). Contate com um polipeptídeo de acordo com a invenção (100ng/ml_-100 pg/mL, preferivelmente 200 ng/mL a 25 pg/mL) durante 3-7 dia o s deve resultar em pelo menos um dois aumento de prega na proliferação das células T. Contato tal como descrito acima durante 2 a 3 horas deve resultar em ativação das células T, como medido utilizando de ensaios de citoquina padrão nos quais um dois aumento de prega no nível da liberação de citoquina (por exemplo, TNF ou IFN-y) é indicativo da ativação de célula T (ver Coli- gan et al., Protocolos Atuais em Imunologia, volume 1 (1998)). Células T que o foram ativadas em resposta a um polipeptídeo, polinucleotídeo ou APC que expressa o polipeptídeo pode ser CD4+ e/ou CD8+. As células T específicas para a proteína podem ser expandidas usando técnicas padrão. Dentro de modalidades preferidas, as células T são derivadas de um paciente, um doador relacionado ou um doador não relacionado, e ser administrado ao paciente depois de estimulação e expansão.
[00356]Nas composições farmacêuticas de acordo com a invenção, a formu-lação de excipientes farmaceuticamentes aceitávéis e soluções de veículo é bem conhecida para aqueles com habilidade na técnica, como é o desenvolvimento de regimes de tratamento e dosagem adequados para usar as composições particulares aqui descritas em vários regimes de tratamento incluindo, por exemplo, administração oral, parenteral, intravenosa, intranasal, intradérmica, subcutânea, e intramuscular e formulação.
[00357]Em certos Pedidos, as composições farmacêuticas aqui divulgadas podem ser liberadas via a administração oral a um paciente. Como tal, estas compo-sições podem ser formuladas com um diluente inerte ou com um veículo comestível assimilável, ou elas podem ser elvolvidas por uma cápsula de gelatina dura ou macia, ou elas podem ser comprimidos em pílulas, ou elas podem ser incorporadas diretamente com o alimento da dieta prescrita.
[00358]Em certas circunstâncias será desejável liberar as composições far-macêuticas aqui divulgadas parenteralmente, intravenosamente, intramuscularmente, ou até intraperitonealmente como descrito, por exemplo, na Patente dos Estados Unidos N° US. 5.543.158; Patente dos Estados Unidos N° US. 5.641, 515 e Patente dos Estados Unidos N° US. 5.399.363 (cada um especificamente incorporado aqui por referência em sua totalidade). As soluções dos compostos ativos como base livre ou sais farmacologicamente aceitáveis podem ser preparadas na água adequadamente mistura com um tensoativo, tal como hidroxipropilcelulose. As dispersões também pode ser prepararam em glicerol, polietileno glicol líquido, e misturas dos mesmos e em óleos. As condições sob normais do armazenamento e uso, estas preparações contêm um preservativo para prevenir o crescimento de microrganismos.
[00359]As formas farmacêuticas adequadas para o uso injetável incluem so-luções aquosas estéreis ou dispersões e pó estéril da preparação extemporânea de soluções injetáveis estéreis ou dispersões (Patente dos Estados Unidos N° US. 5.466.468, especificamente incorporada aqui por referência em sua totalidade). Em todos os casos a forma deve ser estéril e deve ser fluido até o ponto que existe um fluxo fácil através de seringa. Deve ser estável sob as condições de produção e ar-mazenamento e deve ser conservado contra a ação de contaminação de microrga-nismos, tais como bactérias e fungos. O veículo pode ser um solvente ou meio de dispersão contendo, por exemplo, água, etanol, poliol (por exemplo, glicerol, propile- no glicol, e polietileno glicol líquido, e assim por diante), misturas dos mesmos adequados, óleos de verdura de and/or. A fluidez própria pode ser mantida, por exemplo, pelo uso de um revestimento, tal como lecitina, pela manutenção do tamanho de partícula necessário no caso da dispersão e pelo uso de tensoativos. A prevenção da ação de microrganismos pode ser facilitada por agentes antibacterianos e anti- fúngicos diferentes, por exemplo, parabenos, clorobutanol, fenol, ácido sórbico, time- rosal, e assim por diante. Em muitos casos, será preferível incluir agentes isotônicos, por exemplo, açúcar ou cloreto de sódio. A absorção prolongada das composições injetáveis pode ser ocasionada pelo uso nas composições de agentes que atrasam a absorção, por exemplo, monoestearato de alumínio e gelatina.
[00360]Para a administração parenteral em uma solução aquosa, por exemplo, a solução deve ser adequadamente armazenada em buffer se necessário e o diluente líquido primeiro produziu o isotônico com salina suficiente ou glicose. Estas soluções aquosas particulares são especialmente adequadas para a administração intravenosa, intramuscular, subcutânea e intraperitonial. Nesta conexão, um meio aquoso estéril que pode ser empregado será conhecido daqueles com habilidade na técnica na luz da divulgação presente. Por exemplo, uma dosagem pode ser dissolvida em 1 ml_ do isotônico solução de NaCI e acrescentada a 1000 ml_ de fluido hypodermoclysis ou injetada no sítio proposto da infusão (ver, por exemplo, Remington: a Science e Prática de Farmácia, 20a Edição. O Baltimore, Maryland: Lippincott Williams & Wilkins, 2000). A pouca variação na dosagem ocorrerá necessariamente dependendo da condição do paciente que é tratado. A pessoa responsável pela administração determinará, em todo o caso, a dose apropriada ao paciente individualmente. Além disso, para a administração humana, as preparações devem estar de acordo com a esterilidade, pirogenicidade, e a segurança geral e os padrões de pureza tal como exigido pelo escritório do FDA de padrões biológicos.
[00361]As soluções injetáveis estéreis são preparadas incorporando os compostos ativos na quantidade necessária no solvente apropriado com outros in-gredientes diferentes listados acima, como necessitado, seguido pela esterilização filtrada. Geralmente, as dispersões são preparadas incorporando os ingredientes ativos esterilizados diferentes em um veículo estéril contendo o meio de dispersão básico e outros ingredientes necessários dos listados acima. Em caso do pó estéril da preparação de soluções injetáveis estéreis, os métodos preferidos da preparação são técnicas secam o vácuo e congelam a vácuo que produzem um pó do ingrediente ativo mais o ingrediente desejado adicional qualquer de uma solução anteriormente filtrada de maneira estéril do mesmo.
[00362]As composições aqui divulgadas podem ser formuladas em uma forma neutra ou forma de sal. Sais farmaceuticamentes aceitáveis, incluem os sais de adição ácida (formado com os grupos de amina livres da proteína) e que são formados com ácidos inorgânicos tal como, por exemplo, ácidos clorídricos ou fosfóricos, ou tais ácidos orgânicos como acético, oxálico, tartárico, mandelic, e assim por diante. Os sais formados com os grupos de carboxila livres também podem ser derivados de bases inorgânicas tal como, por exemplo, sódio, potássio, amónio, cálcio, ou hidróxido férrico, e tais bases orgânicas como isopropilamina, trimetilamina, histidina, procaína e assim por diante. Sobre a formulação, as soluções serão administradas em uma maneira compatível com a formulação de dosagem e em tal quantidade como é terapeuticamente eficaz. As formulações são facilmente administradas em várias formas de dosagem, tais como soluções injetáveis, cápsulas de liberação da droga, e assim por diante. Tal como aqui utilizado, "o veículo" inclui qualquer e todos os solventes, meios de dispersão, veículos, revestimentos, diluentes, agentes anti- bacterianos e antifúngicos, isotônicos e agentes de atraso de absorsão, tampões, soluções de veículo, suspensões, colóides, e assim por diante. O uso de tal meio e agentes de substâncias ativas farmacêuticas é bem conhecido na técnica. Exceto à medida que meio convencional qualquer ou agente é incompatível com o ingrediente ativo, his uso nas composições terapêuticas é contemplado. Os ingredientes ativos suplementares também podem ser incorporados nas composições.
[00363]A frase "farmaceuticamente aceitável" refere-se a entidades moleculares e composições que não produzem uma reação desfavorável alérgica ou semelhante quando administrado para um ser humano. A preparação de uma composição aquosa contendo uma proteína como um ingrediente ativo é bem entendida na técnica. Tipicamente, tais composições são preparadas como injectables, como soluções líquidas ou suspensões; formas sólidas adequadas para a solução em, ou suspensão em, o líquido antes da injeção também pode ser preparado. A preparação também pode ser emulsionada.
[00364]Em certas modalidades, as composições farmacêuticas podem ser liberadas por borrifos intranasais, inalação, e/ou outros veículos de liberação de ae-rossol. Os métodos para liberar genes, polinucleotídeos, e composições de peptídio diretamente aos pulmões via o aerossol nasal borrifam foi descrito por exemplo, no Patente dos Estados Unidos N° US. 5.756.353 e Patente dos Estados Unidos N° US. 5.804.212 (cada um especificamente incorporado aqui por referência em sua totali-dade). De mesmo modo, a liberação de drogas usando resinas de micropartícula intranasais (Takenaga et al., 1998) e compostos de lisofosfatidil-glicerol (Patente dos Estados Unidos N° US. 5.725.871, especificamente incorporado aqui pela referência em sua totalidade) são também bem conhecidos nas técnicas farmacêuticas. De mesmo modo, liberação de droga transmucosa na forma de um politetrafluoroetileno suportado em matriz é descrita na Patente dos Estados Unidos N° US. 5.780.045 (especificamente incorporado aqui por referência em sua totalidade).
[00365]Em certas modalidades, a liberação pode ocorrer pelo uso de lipos- somas, nanocapsules, micropartículas, microesferas, partículas de lipídio, vesículas, e assim por diante, para a introdução das composições da presente invenção em células de hospedeiros adequados. Particularmente, as composições da presente invenção podem ser formuladas para a liberação encapsulada em uma partícula de lipídio, um lipossoma, uma vesícula, um nanosphere, uma nanopartícula ou o parecido. A formulação e o uso de tais veículos de liberação podem ser técnicas conhecidas e convencionais usam realizadas.
D. Métodos Diagnósticos e Kits.
[00366]Como observado acima, as composições, os polipeptídeos de fusão e os polinucleotídeos são também úteis como reagentes diagnósticos para detectar e/ou que controla infecção de Mycobacterium tuberculosis em um paciente. Por exemplo, as composições, os polipeptídeos de fusão, e os polinucleotídeos de acordo com a invenção podem ser usados em testes in vitro e in vivo para detectar anticorpos humorais ou imunidade celular mediada contra Mycobacterium tuberculosis para o diagnóstico da infecção, controlando de progressão de doença ou avaliação de teste da cura. Desse modo, em certas modalidades, a invenção fornece antígenos para diagnósticos melhorados para diagnosticar diferencialmente a infecção de tuberculose por Mycobacterium baseada no exame sorológico, em que os antígenos de Mycobacterium usados no diagnóstico são selecionados do grupo consistindo de Rv0164 (SEQ ID NO: 1), Rv0496 (SEQ ID NO: 6), Rv2608 (SEQ ID NO: 26), Rv3020 (SEQ ID NO: 36), Rv3478 (SEQ ID NO: 41), Rv3619 (SEQ ID NO: 46), Rv3620 (SEQ ID NO: 51), RV1738 (SEQ ID NO: 11), Rv1813 (SEQ ID NO: 16), Rv3810 (SEQ ID NO: 56), Rv2389 (SEQ ID NO: 21), Rv2866 (SEQ ID NO: 31), Rv3876 (SEQ ID NO: 61), Rv0054 (SEQ ID NO: 100), Rv0410 (SEQ ID NO: 106), Rv0655 (SEQ ID NO: 112), Rv0831 (SEQ ID NO: 115), Rv1009 (SEQ ID NO: 118), Rv1099 (SEQ ID NO: 121), Rv1240 (SEQ ID NO: 124), Rv1288 (SEQ ID NO: 127), Rv1410 (SEQ ID NO: 130), Rv1569 (SEQ ID NO: 133), Rv1789 (SEQ ID NO: 136), Rv1818 (SEQ ID NO: 139), Rv1860 (SEQ ID NO: 142), Rv1886 (SEQ ID NO: 145), Rv1908 (SEQ ID NO: 148), Rv2220 (SEQ ID NO: 154), Rv2032 (SEQ ID NO: 151), Rv2623 (SEQ ID NO: 160), Rv2875 (SEQ ID NO: 163), Rv3044 (SEQ ID NO: 166), Rv3310 (SEQ ID NO: 169), e Rv3881 (SEQ ID NO: 178), Rv0577 (SEQ ID NO: 184), Rv1626 (SEQ ID NO: 187), Rv0733 (SEQ ID NO: 190), Rv2520 (SEQ ID NO: 193), Rv1253 (SEQ ID NO: 196), Rv1980 (SEQ ID NO: 199), Rv3628 (SEQ ID NO: 202) Rv1884 (SEQ ID NO: 205), Rv3872 (SEQ ID NO: 208), Rv3873 (SEQ ID NO: 211), Rv1511 (SEQ ID NO: 214) e Rv3875 (SEQ ID NO: 292), ou porções imunogênicas ou variantes dos mesmos, em quaisquer combinações e misturas dos mesmos como antígenos separados, ou em construtores genéticos de fusão. Como demonstrado aqui, as combinações dos antígenos diagnósticos divulgados oferecem a sensibilidade melhorada no teste diagnóstico sorológico.
[00367]Qs métodos diagnósticos e os kits preferivelmente empregam uma combinação de dois ou mais antígenos tal como aqui descrito. Em certas modalidades, será preferido para usar múltiplos antígenos tal como aqui descrito, por exemplo, três ou mais, quatro ou mais, cinco ou mais, seis ou mais, etc., em um método diagnóstico de acordo com a invenção. Os antígenos podem ser usados essencialmente em qualquer formato de ensaio desejado, por exemplo, como antígenos individuais analisados separadamente, como múltiplos ensaios de antígenos simultaneamente, como os antígenos imobilizaram sobre uma o suporte sólido, tal como uma tabela, ou parecido.
[00368]Em uma modalidade particular, os antígenos diagnósticos usados nos métodos aqui são selecionados do grupo consistindo de Rv0164 (SEQ ID NO: 1), Rv0496 (SEQ ID NO: 6), Rv2608 (SEQ ID NO: 26), Rv3020 (SEQ ID NO: 36), Rv3478 (SEQ ID NO: 41), Rv3619 (SEQ ID NO: 46), Rv3620 (SEQ ID NO: 51), RV1738 (SEQ ID NO: 11), Rv1813 (SEQ ID NO: 16), Rv3810 (SEQ ID NO: 56), Rv2389 (SEQ ID NO: 21), Rv2866 (SEQ ID NO: 31), Rv3876 (SEQ ID NO: 61), Rv0054 (SEQ ID NO: 100), Rv0410 (SEQ ID NO: 106), Rv0655 (SEQ ID NO: 112), Rv0831 (SEQ ID NO: 115), Rv1009 (SEQ ID NO: 118), Rv1099 (SEQ ID NO: 121), Rv1240 (SEQ ID NO: 124), Rv1288 (SEQ ID NO: 127), Rv1410 (SEQ ID NO: 130), Rv1569 (SEQ ID NO: 133), Rv1789 (SEQ ID NO: 136), Rv1818 (SEQ ID NO: 139), Rv1860 (SEQ ID NO: 142), Rv1886 (SEQ ID NO: 145), Rv1908 (SEQ ID NO: 148), Rv2220 (SEQ ID NO: 154), Rv2032 (SEQ ID NO: 151), Rv2623 (SEQ ID NO: 160), Rv2875 (SEQ ID NO: 163), Rv3044 (SEQ ID NO: 166), Rv3310 (SEQ ID NO: 169), e Rv3881 (SEQ ID NO: 178), Rv0577 (SEQ ID NO: 184), Rv1626 (SEQ ID NO: 187), Rv0733 (SEQ ID NO: 190), Rv2520 (SEQ ID NO: 193), Rv1253 (SEQ ID NO: 196), Rv1980 (SEQ ID NO: 199), Rv3628 (SEQ ID NO: 202) Rv1884 (SEQ ID NO: 205), Rv3872 (SEQ ID NO: 208), Rv3873 (SEQ ID NO: 211), Rv1511 (SEQ ID NO: 214) e Rv3875 (SEQ ID NO: 292), ou porções imunogênicas ou variantes dos mesmos, em combinação qualquer do mesmo, misturas como antígenos separados, ou em construtores genéticos de fusão.
[00369]Em uma modalidade, são fornecidos kits de diagnóstico para detectar a infecção de Mycobacterium tuberculosis em uma amostra biológica, compreendendo (a) um polipeptídeo compreendendo pelo menos uma porção imunogênica de um antígeno ou polipeptídeo de fusão aqui descrito, e (b) um reagente de detecção. Em outra modalidade, são fornecidos kits de diagnóstico para detectar a infecção de Mycobacterium tuberculosis em uma amostra biológica, compreendendo (a) um anti-corpo ou antígeno fragmento obrigatório do mesmo que é específico para um o poli-peptídeo compreendendo pelo menos uma porção imunogênica de um antígeno ou polipeptídeo de fusão aqui descrito, e (b) um reagente de detecção.
[00370]Em outra modalidade, são fornecidos os métodos para detecção da presença da infecção por Mycobacterium tuberculosis em uma amostra biológica, compreendendo (a) contactar uma amostra biológica com um anticorpo monoclonal que se liga a um antígeno ou polipeptídeo de fusão aqui descrito; e (b) detectar na amostra biológica a presença de proteínas de Mycobacterium tuberculosis que ligam ao anticorpo monoclonal.
[00371]Em ainda outra modalidade, são fornecidos os métodos para detecção da infecção por Mycobacterium tuberculosis em uma amostra biológica, compreendendo (a) contactar a amostra biológica com uma combinação de antígeno ou polipeptídeo de fusão tal como aqui descrito e (b) detectar na amostra biológica a presença de anticorpos e/ou de células T que se ligam ao mesmo.
[00372]Existem vários formatos de ensaio conhecidos daqueles com habilidade normal na técnica para usar o antígeno purificado ou o polipeptídeo de fusão para detectar anticorpos em uma amostra. Ver, por exemplo, Harlow e Travessa, Antibodies: A Laboratory Manual, Cold Spring Harbor Laboratory, 1988. Em uma modalidade, o ensaio envolve o uso do polipeptídeo imobilizado sobre um suporte sólido para ligar a e remover o anticorpo da amostra. O anticorpo ligado então pode ser detectado usando um reagente de detecção que liga ao complexo de anticor- po/peptídio e contém um grupo de repórter detectável. Os reagentes de detecção adequados incluem anticorpos que ligam ao anticorpo/polipeptídeo o polipeptídeo complexo e livre marcador com um grupo de repórter (por exemplo, em um ensaio semi-competitivo). Alternativamente, um ensaio competitivo pode ser utilizado, em que um anticorpo que liga ao polipeptídeo é marcador com um grupo de repórter e permitido ligar ao antígeno imobilizado depois da incubação do antígeno com a amostra. O ponto até o qual os componentes da amostra inibição a ligação do anticorpo marcado ao polipeptídeo é indicativo da reatividade da amostra com o polipeptídeo imobilizado.
[00373]O suporte sólido pode ser material sólido qualquer conhecido daqueles com habilidade normal na técnica à qual o antígeno pode ser anexado. Por exemplo, o suporte sólido pode ser um teste poço em uma microplaca ou um nitro- cellulose ou outra membrana adequada. Altemativamente, o suporte pode ser uma conta ou disco, tal como vidro, fibra de vidro, látex ou um material plástico, tal como poliestireno ou cloreto de polivinila. O suporte também pode ser uma partícula magnética ou um sensor de fibra ótica, tal como os divulgados, por exemplo, na Patente dos Estados Unidos N° US 5.359.681.
[00374]O polipeptídeo pode estar ligado ao suporte sólido usando qualquer de várias técnicas conhecidas e disponíveis na técnica. O termo "ligado" refere-se à associação não covalente de ambos, tal como adsorção, e anexação covalente (que pode ser uma ligação direta entre o antígeno e grupos funcionais do suporte ou pode ser uma ligação por meio de um agente de reticulação). Ligar por adsorção ao poço em uma microplaca ou a uma membrana é preferido. Em tais casos, a adsorção pode ser atingida contatando com o polipeptídeo, em um tampão adequado, com o suporte sólido período de tempo para um adequado.
[00375]Em certas modalidades, o ensaio diagnóstico empregado é o ensaio imunosorbent ligado de uma enzima (ELISA). Este ensaio pode ser realizado conta-tando primeiro com um antígeno de polipeptídeo que foi imobilizado sobre um suporte sólido, comumente o poço de uma microplaca, com a amostra, de tal modo que o polipeptídeo dos anticorpos dentro da amostra são deixados ligar ao polipeptídeo imobilizado. A amostra não ligada então é removida do polipeptídeo imobilizado e um reagente de detecção capaz de ligação ao complexo polipeptídeo e anticorpo imobilizado é acrescentado. A quantidade do reagente de detecção que permanece ligado ao suporte sólido é então determinada usando um método apropriado para o reagente de detecção específico.
[00376]Uma vez que o polipeptídeo é imobilizado no suporte, os sítios de li-gação de proteína restantes no suporte são tipicamente bloqueados. Agente bloque- ador adequado qualquer conhecido àqueles de habilidade normal na técnica, tais como albumina de soro bovino ou Tween 20® (Sigma Chemical Co, St. Louis, Mis- souri). O polipeptídeo imobilizado então é incubado com a amostra, e permite-se que o anticorpo (se presente na amostra) se ligue ao antígeno. A amostra pode ser diluída com um diluente adequado, tal como tampão de fosfato salino (PBS) antes da incubação. Em geral, um tempo de contato apropriado (isto é, o tempo de incubação) é um período de tempo que é suficiente para detectar a presença do anticorpo ao Mycobacterium tuberculosis dentro de uma amostra infectada. Preferivelmente, o tempo de contato é suficiente para atingir um nível da ligação que é pelo menos de 95 % atingido no equilíbrio entre o anticorpo ligado e não ligado. Aqueles com habilidade normal na técnica reconhecerão que o tempo necessário para atingir o equilíbrio pode ser prontamente determinado analisando o nível da ligação que ocorre durante o período do tempo. Na temperatura ambiente, um tempo de incubação de aproximadamente 30 minutos é geralmente suficiente.
[00377]A amostra não ligada então pode ser removida lavando o suporte sólido com um tampão apropriado, tal com PBS contendo Tween20® a 0,1 %. O reagente de detecção então pode ser acrescentado ao suporte sólido. Um reagente de detecção apropriado é qualquer composto que liga ao complexo de polipeptídeo do anticorpo imobilizado e isto pode ser detectado por qualquer de vário significa conhecido àqueles na técnica. O reagente de detecção geralmente contém um agente de ligação (tal como, por exemplo, proteína A, proteína G, imunoglobulina, lecitina ou antígeno livre) conjugado a um grupo de repórter. Os grupos de repórter ilustrativos incluem enzimas (tais como peroxidase de rábano silvestre), substratos, cofatores, inibidores, corantes, radionuclídios, grupos luminescentes, grupos fluorescentes e biotina. A conjugação do agente de ligação ao grupo de repórter pode ser atingida usando métodos padrão conhecidos daqueles com habilidade normal na técnica.
[00378]O reagente de detecção então é incubado com o complexo de poli-peptídeo do anticorpo imobilizado de um período de tempo suficiente para detectar o anticorpo ligado. Um período de tempo apropriado pode ser geralmente determinado das instruções do fabricante ou analisando o nível da ligação que ocorre durante o período do tempo. O reagente de detecção não ligado então é removido e o reagente de detecção ligado é detectado usando o grupo de repórter. O método empregado para detectar o grupo de repórter depende na natureza do grupo de repórter. Para grupos radioativos, a contagem de cintilação ou os métodos autoradiographic são geralmente apropriados. Os métodos de Spectroscopic podem ser usados para detectar corantes, grupos luminescentes e grupos fluorescentes. A biotina pode ser detectada usando avidin, ligada a um grupo de repórter diferente (comumente um grupo radioativo ou fluorescente ou uma enzima). O repórter de enzima grupos pode ser geralmente detectado pela adição do substrato (geralmente para um período de tempo específico), seguido por análise espectroscópica ou outra análise dos produtos da reação.
[00379]Para determinar a presença ou a ausência de anticorpos de Myco-bacterium tuberculosis em uma amostra, o sinal detectado do grupo de repórter que permanece ligado ao suporte sólido é geralmente comparado com um sinal que equivale a um valor pré-determinado de cut-off. Este valor de cut-offé preferivelmente a média das médias do sinal obtido quando o antígeno imobilizado é incubado com amostras de um paciente não infectado. Em geral, uma amostra que gera um sinal que é três desvios padrão acima da média é considerado positivo para anticorpos de Mycobacterium tuberculosis e para a infecção de Mycobacterium tuberculosis. Em outra modalidade, o valor do cut-off é determinado usando uma Curva de Operador de Receptor, de acordo com o método de Sackett et al., Clinical Epidemiology: A Basic Science for Clinicai Medicine, p. 106-7 (Little Brown and Co, 1985). Resumidamente, nesta modalidade, o valor do cut-off pode ser determinado de um lote de pares de taxas de positivos verdadeiros (isto é, sensibilidade) e taxas falsos positivos (100 % de especificidade) que equivalem a cada valor do cut-off possível do resultado de experiência diagnóstico. O valor do cut-off no lote que é o muito próximo à esquina canhota superior (isto é, o valor que cerca a maior área) é o valor do cut-off bem mais exato, e uma amostra que gera um sinal que é mais alto do que o valor do cut-off determinado por este método pode considerar-se positivo. Alternati- vamente, o valor do cut-off pode ser deslocado à esquerda ao longo do lote, para minimizar a taxa de falso positivo, ou à direita, minimizar a taxa de falso negativo. Em geral, uma amostra que gera um sinal que é mais alto do que o valor do cut-off determinado por este método considera-se positiva para a infecção de Mycobacterium tuberculosis.
[00380]Em outra modalidade, um ensaio diagnóstico pode ser realizado através de um fluxo ou um teste em forma de tira, em que o polipeptídeo de fusão ou antígeno é imobilizado sobre uma membrana, tal como nitrocelulose. No teste através de fluxo, os anticorpos dentro da amostra se ligam ao polipeptídeo imobilizado enquanto a amostra passa pela membrana. Um reagente de detecção (por exemplo, proteína A-ouro coloidal) então se liga ao complexo de polipeptídeo e anticorpo enquanto a solução contendo os reagentes de detecção flui através da membrana. A detecção do reagente de detecção ligado então pode ser realizada tal como descrito acima. No formato do teste de tira, uma extremidade da membrana à qual o polipeptídeo está ligado é imersa em uma solução contendo a amostra. A amostra migra ao longo da membrana por uma região contendo reagente de detecção e à área do polipeptídeo imobilizado. A concentração do reagente de detecção no polipeptídeo indica a presença de anticorpos de Mycobacterium tuberculosis na amostra. Tal teste pode ser tipicamente realizado com uma quantidade muito pequena (por exemplo, uma gota) de soro ou sangue do paciente.
[00381]Em ainda outra modalidade, são fornecidos os métodos para detecção de Mycobacterium tuberculosis de mostras biológicas utilizando anticorpos (que podem ser policlonais ou monoclonais) e/ou as células T específicas para um ou mais antígenos, polipeptídeos de fusão e/ou porções imunogênicas de acordo com a invenção.
[00382]Todas as publicações e os pedidos Patentes citados neste Relatório Descritivo são aqui incorporados pela referência como se cada publicação individual ou Pedido de Patente fosse especificamente e individualmente indicado para ser incorporada pela referência.
[00383]Embora a invenção precedente tenha sido descrita em alguns detalhes por meio de ilustração e exemplo com objetivos da esclarecer da compreensão, é prontamente evidente para a pessoa com habilidades normais na técnica à luz dos ensinamentos da presente invenção, que certas modificações e alterações podem ser feitas à mesma sem fugir do espírito ou do alcance das reivindicações anexadas. Os seguintes exemplos são fornecidos por meio da ilustração somente e não por meio da limitação. Aqueles com habilidade na técnica reconhecerão prontamente vários parâmetros não críticos que podem ser modificados ou modificados para produzir resultados essencialmente semelhantes.
[00384]As várias modalidades descritas acima podem ser combinadas para fornecer modalidades adicionais. Todas das Patentes dos Estados Unidos, as publi-cações dos Pedidos de Patente dos Estados Unidos, os Pedidos Patentes dos Estados Unidos, as Patentes estrangeiras, os Pedidos de Patentes estrangeiras e as Pu-blicações que não são Patentes referidas nesse Relatório Descritivo e/ou listada na Folha de Dados do Pedido, são incorporadas aqui pela referência, nhis conjunto. Os aspectos das modalidades podem ser modificados, se necessário para empregar conceitos das diversas Patentes, Pedidos e Publicações para fornecer ainda modali-dades adicionais.
EXEMPLOS.
EXEMPLO 1.
CLONAGEM E EXPRESSÃO DA RV0164 RECOMBINANTE.
[00385]Usando DNA genômico H37Rv como padrão, Rv0164 foi amplificada por PCR utilizando os iniciadores apresentados nas SEQ ID NO: 4 e 5, abaixo: Iniciador 5’- Rv0164-5his-Ndel: TAGGATCCCATATGACGGCAATCTCGTGCTCAC (SEQ ID NO: 4). Iniciador 3’- Rv0164-3Hindlll: TAGAATTCAAGCTTTTAGCTGGCCGCCAGCTGCTC (SEQ ID NO: 5).
[00386]As seguintes condições de amplificação são usadas: 94 °C 0.5 minutos, 55 °C 0,5 minutos, 68 °C 1 min de 30 ciclos para fornecer o produto apresentado na SEQ ID NO: 2. O produto do PCR foi digerido com Ndel / Hindlll e clonado na pET 28a. O plasmídio contendo o gene Rv0164 foi transformado no hospedeiro de expressão e Rosetta2 pLysS. As culturas foram cultivadas em frasco com agitação em 37 °C em 2x meio YT complementado com 34 mg/L de cloranfenicol, 35 mg/mL de canamicina a uma ODeoo = 0,5 a 0,6 e induzido com 1 mM IPTG por 3 a 4 h. A pasta de células foi peletizada em 10000 x g e guardada a -20 °C. Depois da lise de 1 L da solução da solução de indução por ultrassom e clarificação do sobrenadante, a proteína Rv0164 permaneceu na fração insolúvel. Esta fração então foi lavada 2X em detergente CHAPS a 1 %, 20 mM de Tris HCI em pH 8,0, e depois solubilizada em uréia a 8M. A purificação foi atingida usando 2 círculos da cromatografia de afinidade Ni-NTA (Qiagen) que sob condições de desnaturação e a proteína Rv0164 foi eluída utilizando imidazol a 300 mM. Depois da análise de SDS-PAGE, as frações contendo a proteína purificada são dializadas contra 10 mM de Tris em pH 8,0. A concentração de proteína foi determinada pelo teste de Bradford e os níveis de en- dotoxinas residuais são determinados pelo teste de Llimuluis Amoebcyte. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 3.
EXEMPLO 2.
CLONAGEM E EXPRESSÃO DA RV0496 RECOMBINANTE.
[00387]Usando DNA genômico H37Rv como padrão, Rv0496 foi amplificada por PCR utilizando os seguintes iniciadores: 5’-RvO496-5his-Ndel TAGGATCCCATATGGTCGATGCCCACCGCGGC (SEQ ID NO: 9). 3’-RvO496-3Hindlll TAGAATTCAAGCTTTCATGGTTTGCTGCCTCTCGA (SEQ ID NO: 10).
[00388]A amplificação foi realizada sob as seguintes condições: 94 °C 0,5 minutos, 55 °C 0,5 minutos, 68 °C 2 min de 30 ciclos, para fornecer o produto apre-sentado na SEQ ID NO: 7. O produto do PCR foi digerido com Ndel / Hindlll e clona- do em pET28a. Rv0496 foi transformada nos hospedeiros de expressão e Rosetta2 plysS. Depois da lise de 1 L da solução de indução, ele entrou no corpo de inclusão. A Ni-NTA foi realizada duas vezes sob condições de desnaturação, depois dializada contra Tris a 10 mM pH 10. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 8.
EXEMPLO 3.
CLONAGEM E EXPRESSÃO DA RV1738 RECOMBINANTE.
[00389]Usando DNA genômico H37Rv como padrão, Rv1738 foi amplificada por PCR utilizando os seguintes iniciadores: 5’-Rv1738-5his-Ndel CAATTACATATGCATCACCATCACCATCACATGTGCGGCGACCAGTCGG AT (SEQ ID NO: 14) 3’-Rv1738-3EcoRI CAATTAGAATTCTCAATACAACAATCGCGCCGG (SEQ ID NO: 15)
[00390]A amplificação foi realizada usando as seguintes condições: 95 °C 1 minuto, 58 °C 1 minuto, 72 °C 1 min de 35 ciclos, para fornecer o produto apresentado como SEQ ID NO: 12. O produto do PCR foi digerido com Ndel / EcoRI e clonado na pET 17b. Rv1738 foi transformada nos hospedeiros de expressão BL-21 plysE e plysS. Depois da lise de 1 L da solução de indução, a proteína permaneceu no so- brenadante solúvel. A Ni-NTA foi realizada sob condições de desnaturação, depois dializada contra 10 mM de Tris em pH 8,0. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 13.
EXEMPLO 4.
CLONAGEM E EXPRESSÃO DA RV1813 RECOMBINANTE.
[00391 ]Usando DNA genômico H37Rv como padrão, a Rv1813 foi amplificada por PCR utilizando os seguintes iniciadores: 5’-Rv1813-5his33-Ndel- CAATTACATATGCATCACCATCACCATCACCATCTCGCCAACGGtTT- CGATG (SEQ ID NO: 19). 3’-Rv1813-3EcoRI- CAATTAGAATTCTTAGTTGCACGCCCAGTTGAC (SEQ ID NO: 20).
[00392]A amplificação foi realizada usando as seguintes condições 95 °C 1 minuto, 58 °C 1 minuto, 72 °C 1 min de 35 ciclos, para fornecer o produto apresentado na SEQ ID NO: 17. O produto do PCR foi digerido com Ndel / EcoRI e clonado na pET 17b. Rv1813 foi transformado em BL-21plysE de hospedeiros de expressão e Rosetta plysS. Depois da lise de 1 L da solução de indução, a proteína entrou no corpo de inclusão. Ni-NTA foi realizada sob condições de desnaturação, depois dializada contra 10 mM de Tris em pH 8,0. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 18.
EXEMPLO 5.
CLONAGEM E EXPRESSÃO DA RV2389 RECOMBINANTE (RPF-D).
[00393]Usando DNA genômico H37Rv como padrão, Rv2389 foi amplificada por PCR utilizando os seguintes iniciadores: 5’-Rv2389-5his50-Ndel- CAATTACATATGCATCACCATCACCATCACGACGACATCGATTGGGACG CC (SEQ ID NO: 24). 3’-Rv2389-3EcoRI- CAATTAGAATTCTCAATCGTCCCTGCTCCCCGA (SEQ ID NO: 25).
[00394]A amplificação foi realizada sob as seguintes condições: 95 °C 1 minuto, 58 °C 1 minuto, 72 °C 1 min de 35 ciclos, para fornecer o produto apresentado na SEQ ID NO: 22. O produto do PCR foi digerido com Ndel / EcoRI e clonado na pET 17b (o construtor pET começa em aa49). Rv2389 foi transformada nos hospedeiros de expressão BL-21 plysE e Rosetta plysS. Depois da lise de 1 L da solução de indução, a proteína permaneceu na fração solúvel. Ni-NTA foi realizada sob condições de desnaturação, depois dializada contra 10 mM de Tris em pH 8,0. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 23.
EXEMPLO 6.
CLONAGEM E EXPRESSÃO DA RV2608 RECOMBINANTE.
[00395]Usando DNA genômico H37Rv como padrão, Rv2608 foi amplificada por PCR utilizando os seguintes iniciadores: 5’-Rv2608-5-Ndel- TAGGATCCCATATGAATTTCGCCGTTTTGCCG (SEQ ID NO: 29) 3’-Rv2608-3-Hindlll- TAGAATTCAAGCTTTTAGAAAAGTCGGGGTAGCGCC (SEQ ID NO: 30)
[00396]A amplificação foi realizada usando as seguintes condições 94 °C 0,5 minutos, 55 °C 0,5 minutos, 68 °C 2 min de 30 ciclos, para fornecer o produto apresentado na SEQ ID NO: 27. O produto do PCR purificado em gel foi digerido com Ndel / Hindlll e clonado no vetor de expressão pET28a (Clonetech) (o construtor pET começa no aminoácido 1). Rv2608 foi transformada nos hospedeiros de expressão e Rosetta2 pLysS. As culturas foram cultivadas em frasco com agitação em 37 °C em 2x meio de YT complementado com 34 mg/L cloranfenicol, 35 mg/mL de canamicina a um ODeoo = 0,5 a 0,6 e induzido com 1 mM IPTG por 3 a 4 h. A pasta de células foi peletizada em 10000 x g e guardada a -20 °C. Depois da lise de 1 L da solução da solução de indução por ultrassom e clarificação do sobrenadante, a proteína Rv2608 permaneceu na fração insolúvel. Esta fração então foi lavada 2X em detergente CHAPS a 1 %, Tris HCI a 10 mM pH 8,0, e depois solubilizada em uréia a 8M. A purificação foi realizada usando cromatografia de afinidade de Ni-NTA (Qiagen) 2X sob condições de desnaturação e a proteína Rv2608 foi eluída usando imidazol a 300 mM. Depois da análise de SDS-PAGE, as frações contendo a proteína purificada são dializadas contra 10 mM de Tris em pH 8,0. A concentração de proteína foi determinada pelo ensaio de BCA e os níveis de endotoxinas residuais são determinado pelo teste de Llimuluis Amoebcyte. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 28.
EXEMPLO 7.
CLONAGEM E EXPRESSÃO DA RV2866 RECOMBINANTE.
[00397]Rv2866 foi amplificada do padrão genomic por PCR, usando os se-guintes iniciadores: 5’-Rv2866-5Ndel- CAATTACATATGCCTTCCACCGTGCCCTTCACC (SEQ ID NO: 32). 3’-Rv2866-3Hindlll- CAATTAAAGCTTCTATCGGCGGTAGATGTCCGCGCG-(SEQ ID NO: 33).
[00398]As seguintes condições de amplificação são usadas: 94 °C para 0,5 minutos, 66 °C para 0,5 min., 68 °C para 1,50 minutos, 35 ciclos), para fornecer o produto apresentado na SEQ ID NO: 34. O produto foi digerido com Ndel / Hindlll e clonado no vetor pET28.a. Rv2866 foi expressado pela cepa de hospedeiro BL-21 plysS. A pelota e o sobrenadante são ligados com a resina Ni sob condições de desnaturação. A diálise foi realizada em Tris 20 mM pH 6,0. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 35.
EXEMPLO 8.
[00399]Clonagem e expressão da Rv3020 Recombinante Usando H37 ge- nomic DNA como padrão, Rv3020 foi amplificada por PCR utilizando os seguintes iniciadores: 5’-Rv3020-5his-Ndel- TAGGATCCCATATGAGTTTGTTGGATGCCCATAT (SEQ ID NO: 39) 3-Rv3020-3Hindlll- TAGAATTCAAGCTTTTAAAACCCGGTGTAGCTGGAC (SEQ ID NO: 40)
[00400]As seguintes condições de amplificação são empregado: 94 °C 0,5 minutos, 55 °C 0,5 minutos, 68 °C 1 minuto de 30 ciclos, produzindo o produto apresentado na SEQ ID NO: 37. O produto do PCR foi digerido com Ndel / Hindlll e clonado na pET 28a. O plasm ídio contendo o gene Rv3020 foi transformado no hospedeiro de expressão e Rosetta2 pLysS. As culturas foram cultivadas em frasco com agitação em 37 °C em 2x meio de YT complementado com 34 mg/L cloranfenicol, 35 mg/mL de canamicina a um ODeoo = 0,5 a 0,6 e induzido com 1 mM IPTG por 3 a 4 h. A pasta de células foi peletizada em 10000 x g e guardada a -20 °C. Depois da lise de 1 L da solução da solução de indução por ultrassom e clarificação do sobre- nadante, a proteína Rv3020 permaneceu na fração insolúvel. Esta fração então foi lavada 2X em detergente CHAPS a 1 %, 20 mM de Tris HCI em pH 8,0, e depois solubilizada em uréia a 8M. A purificação foi realizada usando cromatografia de afinidade de Ni-NTA (Qiagen) que sob condições de desnaturação e a proteína Rv3020 foi eluída usando 250 Imidazol de mm. Depois da análise de SDS-PAGE, as frações contendo a proteína purificada são dializadas contra 10 mM de Tris em pH 8,0. A concentração de proteína foi determinada pelo teste de Bradford e os níveis de endotoxinas residuais são determinado pelo teste de Llimuluis Amoebcyte. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 38.
EXEMPLO 9
CLONAGEM E EXPRESSÃO DA RV3478 RECOMBINANTE
[00401 ]Usando DNA genômico H37Rv como padrão, Rv3478 foi amplificada usando os seguintes iniciadores: 5’-Rv3478-5his-Ndel- TAGGATCCCATATGGTGGATTTCGGGGCGTTAC (SEQ ID NO: 44) 3-Rv3478-3Hindlll- TAGAATTCAAGCTTCTATCCGGCGGCCGGTGTGCG (SEQ ID NO: 45)
[00402]Rv3478 foi amplificada usando reação de cadeia de polimerase (PCR) com as seguintes condições 94 °C 0,5 minutos, 55 0,5 minutos °C, 68 °C 2 minutos de 30 ciclos. O produto do PCR purificado em gel (SEQ ID NO: 42) foi digerido com Ndel / Hindlll e clonado no vetor de expressão pET28a (Clonetech). Rv3478 foi transformada nos hospedeiros de expressão e Rosetta2 pLysS. As culturas foram cultivadas em frasco com agitação em 37 °C em 2x meio de YT complementado com 34 mg/L cloranfenicol, 35 mg/mL de canamicina a um ODΘOO = 0,5 a 0,6 e induzido com 1 mM IPTG por 3 a 4 h. A pasta de células foi peletizada em 10000 x g e guardou em - 20 °C. Depois da lise de 1 L da solução da solução de indução por ultrassom e clarificação do sobrenadante, a proteína Rv3478 permaneceu na fração insolúvel. Esta fração então foi lavada 2X em detergente CHAPS a 1 %, Tris HCI a 10 mM pH 8,0, e depois solubilizada em uréia a 8M. A purificação foi feita usando cro- matografia de afinidade de Ni-NTA (Qiagen) 2X sob condições de desnaturação e a proteína Rv3478 foi eluída usando imidazol a 300 mM. Depois da análise de SDS- PAGE, as frações contendo a proteína purificada são dializadas contra 10 mM de Tris em pH 8,0. A concentração de proteína foi determinada pelo ensaio de BCA e os níveis de endotoxinas residuais são determinado pelo teste de Llimuluis Amoeb- cyte. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 43. EXEMPLO 10 CLONAGEM E EXPRESSÃO DA RV3619 RECOMBINANTE Usando DNA genômico H37Rv como padrão, Rv3619 foi amplificada usando os seguintes inicia-dores. 5’-Rv3619-5his-Ndel- TAGGATCCCATATGACCATCAACTATCAATTCG (SEQ ID NO: 49) 3 - Rv3619-3Hindlll- TAGAATTCAAGCTTTTAGGCCCAGCTGGAGCCGAC (SEQ ID NO: 50)
[00403]Rv3619 foi amplificada usando reação de cadeia de polimerase (PCR) com as seguintes condições 94 °C 0.5min., 55 °C 0,5 minutos, 68 °C 1 minuto de 30 ciclos. O produto do PCR purificado em gel (SEQ ID NO: 47) foi digerido com Ndel / Hindlll e clonado no vetor de expressão pET28a (Clonetech).
[00404]Rv3619 foi transformada nos hospedeiros de expressão e Rosetta2 pLysS. As culturas foram cultivadas em frasco com agitação em 37 °C em 2x meio de YT complementado com 34 mg/L cloranfenicol, 35 mg/mL de canamicina a um ODeoo = 0,5 a 0,6 e induzido com 1 mM IPTG por 3 a 4 h. A pasta de células foi pele- tizada em 10000 x g e guardada a -20 °C. Depois da lise de 1 L da solução da solução de indução por ultrassom e clarificação do sobrenadante, a proteína Rv3619 permaneceu na fração insolúvel. Esta fração então foi lavada 2X em detergente CHAPS a 1 %, Tris HCI a 10 mM pH 8,0, e depois solubilizada em uréia a 8M. A purificação foi realizada usando cromatografia de afinidade de Ni-NTA (Qiagen) que sob condições de desnaturação e a proteína Rv3619 foi eluída utilizando imidazol a 300 mM. Depois da análise de SDS-PAGE, as frações contendo a proteína purificada são dializadas contra 10 mM de Tris em pH 8,0. A concentração de proteína foi determinada pelo teste de Bradford e os níveis de endotoxinas residuais são determinado pelo teste de Llimuluis Amoebcyte. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 48. EXEMPLO 11 CLONAGEM E EXPRESSÃO DA RV3620 RECOMBINANTE Usando DNA genômico H37Rv como padrão, Rv3620 foi amplificada por PCR utilizando os seguintes iniciadores: 5’-Rv3620-5his-Ndel- TAGGATCCCATATGACCTCGCGTTTTATGACG (SEQ ID NO: 54) 3 - Rv3620-3Hindlll- TAGAATTCAAGCTTTCAGCTGCTGAGGATCTGCTG (SEQ ID NO: 55)
[00405]Rv3620 foi amplificada por PCR com condições 94 °C 0.5min., 55 °C 0,5 minutos, 68 °C 1 minuto de 30 ciclos. O produto do PCR (SEQ ID NO: 52) foi digerido com Ndel / Hindlll e clonado em pET28a. Rv3620 foi transformado no hospedeiro de expressão Rosetta2 plysS. Depois da lise de 1 L da solução de indução, a proteína entrou no corpo de inclusão. Ni-NTA foi realizada sob condições de desnaturação, antígeno logo purificado dializadas contra 20 pH de Tris de mm 8.0, 50 mM NaCI. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 53. EXEMPLO 12 CLONAGEM E EXPRESSÃO DA Rv3810 RECOMBINANTE Usando DNA genômico H37Rv como padrão, Rv3810 foi amplificada por PCR utilizando os seguintes iniciadores: 5’-Rv3810-5his23-Ndel- CAATTACATATGCATCACCATCACCATCACAGTCCTTGTGCAT ATTTTCTTGTC (SEQ ID NO: 59) 3’-Rv3810-3Xhol- CAATTACTCGAGTTAGGCGACCGGCACGGTGATTGG (SEQ ID NO: 60)
[00406]Rv3810 foi amplificada por PCR com condições 95 °C 1 minuto, 58 °C 1 minuto, 72 °C 1.5 minutos de 35 ciclos. O produto do PCR (SEQ ID NO: 57) foi digerido com Ndel / Xhol e clonado na pET 17b (o construtor pET começa no amino- ácido 23). Rv3810 foi transformada nos hospedeiros de expressão BL-21 plysE e Rosetta plysS. Depois da lise de 1 L da solução de indução, a proteína entrou no corpo de inclusão. Ni-NTA foi realizada sob condições de desnaturação, depois diali- zada contra 10 mM de Tris em pH 8,0. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 58. EXEMPLO 13 CLONAGEM E A EXPRESSÃO DA RV3876 RECOMBINANTE o Rv3876 foi amplificada por PCR do DNA genomic usando os seguintes ini-ciadores de amplificação: Rv3876F-Nde-5': GATCCCATGGGCATATGGCGGCCGACTACGAC (SEQ ID NO: 64) Rv3876R-EcorRI-3': GTCAGAATTCTCAACGACGTCCAGCCCT (SEQ ID NO: 65)
[00407]A amplificação foi realizada usando as seguintes condições: 94 °C de 30 s, 55 °C de 30 s, 72 °C 2 minutos de 30 ciclos. O produto do PCR foi ligam no vetor lançador pGemT. Os clones seguros são identificado na libra agar-x-gal placas pela seleção azul/branca. O produto genético Rv3876 foi digerido com Ndel / EcoRI e clonado na pET 28a. Rv3876c foi transformado no hospedeiro de expressão BL-21 (DE3) plysS. Depois da lise de 1 L da solução de indução, a proteína permaneceu na fração insolúvel. Ni-NTA foi realizada sob condições de desnaturação, depois dia- lizada contra 20 pH de Tris de mm 8.0. A sequência de aminoácidos da proteína re-combinante é apresentada na SEQ ID NO: 63. EXEMPLO 14 CLONAGEM E A EXPRESSÃO DA PROTEÍNA DE FUSÃO RECOMBINANTE MTB36F.1 que os seguintes iniciadores são usadas na construção da fusão constrói Mtb36f.1: 5’-Rv2389-5Ndel50- CAATTACATATGGACGACATCGATTGGGACGCC (SEQ ID NO: 68) 3-Rv2389-3Saclgo- CAATTAGAGCTCATCGTCCCTGCTCCCCGAACA (SEQ ID NO: 69) 5’-Rv3810-5Sacl23- CAATTAGAGCTCAGTCCTTGTG] CATATTTTCTTG (SEQ ID NO: 70) 3-Rv3810-3Hindlll-Kpnl- CAATTAAAGCTTTTAGGTACCGGCGACCGGCACGGTGATTG G (SEQ ID NO: 71)
[00408]Usando o DNA de plasmidio anteriormente clonado de Rv2389 e Rv3810, os componentes Mtb36f.1 são o utilizando amplificado por PCR das seguintes condições: 94 °C de 30 s, 58 °C 3Ose α, 68 °C 1 minuto de 35 ciclos. Os 5' Rv2389 PCR produto foram digeridos com Ndel I Triste e clonado no animal 28a. Os 3' Rv3810 PCR produto foram digeridos com Sad I Hindlll e clonaram-se no Rv2389 contendo animal 28a construtor. Mtb36f.1 (SEQ ID NO: 66) foi transformado no hospedeiro de expressão BL-21 (DE3) plysS. Depois da lise de 1 L da solução de indução, a proteína permaneceu na fração solúvel. Ni-NTA foi realizada condições sob nativas, depois dializada contra 20 pH de Tris de mm 8.0. A sequência de aminoácidos da proteína de fusão recombinante é apresentada na SEQ ID NO: 67. EXEMPLO 15 CLONAGEM E A EXPRESSÃO DA PROTEÍNA DE FUSÃO RECOMBINANTE ID58 no qual os seguintes iniciadores são usadas para clonar a fusão constrói ID58, que compreende os parceiros de fusão derivaram de Mtb Rv1813, Rv3620 e Rv0496 5': Rv1813mat-5Ndel-Kpnl CAATTACATATGGGTACCCATCTCGCCAACGGTTCGATG (SEQ ID NO: 73) 3': Rv1813mat-3Saclgo CAATTAGAGCTCGTTGCACGCCCAGTTGACGAT (SEQ ID NO: 74) 5'; Rv3620-5Sacl CAATTAGAGCTCATGACCTCGCGTTTTATGACG (SEQ ID NO: 75) 3': Rv3620-3Sallgo CAATTAGTCGACGCTGCTGAGGATCTGCTGGGA (SEQ ID NO: 76) 5': RvO496-5Sall CAATTAGTCGACATGGTCGATGCCCACCGCGGC (SEQ ID NO: 77) 3'; RvO496-3Scal-Hindlll CAATTAAAGCTTTTAAGTACTTGGTTTGCTGCCTCTCGATCG (SEQ ID NO: 78) Rv1813 e Rv3620 são amplificadas por PCR do padrão genomic
[00409]DNA (94 °C para 0,5 minutos, 58 °C para 0,5 minutos, 58 °C para 1:5 minutos; 35 ciclos). Rv1813 foi digerido com Ndel/Sacl então clonado no vetor pET28.a. Rv3620 foi digerido com Sacl/Sall então ligam no construtor de Rv1813pET. Rv0496 foi amplificada do padrão de plasmidio por PCR (94 oC de 0:30; 60 oC de 0:30; 68 oC para 1:30; 35 ciclos). O produto foi digerido com a Vela / Hindlll e clonado no vetor pET28.a-Rv1813-3620. O id58-pET28.a tinha mutações de ponto poucas assim o sítio mandou que o mutagênese fosse usado para inserir os ácidos nucleicos corretos. O construtor de fusão ID58 mandou apresentar uma sequência de polinucleotídeo em SEQ ID NO: 72, codificando a proteína de fusão apresentada na SEQ ID NO: 79. ID58 foi expressado no hospedeiro BL-21 plysS (1 L, 2XYT meio de crescimento, 37 oC). A indução foi com 1 mM que IPTG em OD 0.471 e células são colhido em OD 1.36. A pelota de célula foi suspensa no tampão de lise (20 Tris de mm pH8, 100 mM NaCI, 2 mM PMSF) e congelou-se. ID58 forma um corpo de inclusão e foi processado o mesmo como ID83. As frações do fluxo por ligam são dializadas em 20 pH de Tris de mm 8.5. EXEMPLO 16 CLONAGEM E EXPRESSÃO DA PROTEÍNA DE FUSÃO RECOMBINANTE ID69 Os seguintes iniciadores são usadas em para clonagem a fusão constroem ID69, que compreende os parceiros de fusão derivaram de Rv2389, Rv1813, Rv3620 e Rv0496: 5': Rv2389mat-5Ndel CAATTACATATGGACGACATCGATTGGGACGCC (SEQ ID NO: 81) 3VRv2389mat-3Kpnl-Hindlll CAATTAAAGCTTTTAAGTACTTGGTTTGCTGCCTCTCGATCG (SEQ ID NO: 82)
[00410]Rv2389 foi amplificada por PCR do padrão genomic (94 °C para 0,5 minutos, 58 °C para 0,5 minutos, 68 °C para 1.5 minutos; 35 ciclos), digerido com Ndel/Hindlll, e ligam em pET28.a. ID58 - pET28.a vetor foi digerido com Kpnl/Hindlll para retirar-se da inserção. ID58 foi ligam em rv2389-pET28.a o vetor (também digerido com Kpnl/Hindlll). O construtor de fusão mandou apresentar uma sequência de polinucleotídeo em SEQ ID NO: 80, codificando a proteína de fusão apresentada na SEQ ID NO: 83. ID69 foi expressado no hospedeiro BL-21plysS (1 L, 2XYT meio de crescimento, 37 oC). A pelota de célula foi suspensa no tampão de lise (20 Tris de mm pH8, 100 mM NaCI, 2 mM PMSF) e congelou-se. ID69 forma um corpo de inclusão e foi purificado o mesmo como ID83. EXEMPLO 17 CLONAGEM E EXPRESSÃO DA PROTEÍNA DE FUSÃO RECOMBINANTE ID83 Os seguintes iniciadores são usadas em para clonagem a fusão constroem ID83, que compreende parceiros de fusão de Rv1813, Rv3620 e Rv2608: 5': Rv1813mat-5Ndel-Kpnl CAATTACATATGGGTACCCATCTCGCCAACGGTTCGATG (SEQ ID NO: 85) 3': Rv1813mat-3Saclgo CAATTAGAGCTCGTTGCACGCCCAGTTGACGAT (SEQ ID NO: 86) 5': Rv3620-5Sacl CAATTAGAGCTCATGACCTCGCGTTTTATGACG (SEQ ID NO: 87) 3': Rv3620-3Sallgo CAATTAGTCGACGCTGCTGAGGATCTGCTGGGA (SEQ ID NO: 88) 5': Rv2608-5Sall CAATTAGTCGACATGAATTTCGCCGTTTTGCCG (SEQ ID NO: 89) 3': Rv2608-3Scal-Hindlll CAATTAAAGCTTTTAAGTACTGAAAAGTCGGGGTAGCGCCGG (SEQ ID NO: 90) Rv1813 e Rv3620 são amplificadas por PCR do padrão genomic
[00411]DNA (94 °C para 0,5 minutos; 58 °C para 0.5min., 58 °C para 1.5 mi-nutos; 35 ciclos). Rv1813 foi digerido com Ndel/Sacl então clonado no vetor pET28.a. Rv3620 foi digerido com Sacl/Sall então ligam no construtor de Rv1813pET. Rv2608 foi amplificada do padrão de plasmidio por PCR (94 °C para 0,5 minutos, 58 °C para 0,5 minutos, 68 °C para 1.5 minutos; 3 ciclos δ). O produto foi digerido com a Vela I Hindlll e clonado no vetor pET28.a-Rv1813-3620. O construtor de fusão mandou apresentar uma sequência de polinucleotídeo em SEQ ID NO: 84, codificando a proteína de fusão apresentada na SEQ ID NO: 91.
[00412]ID83 foi expressado no hospedeiro BL-21 plysS (2L, 2XYT meio de crescimento, 37 °C). Induzido com 1 mM IPTG em OD 0.77 e colhido em OD 1.93. A pelota de célula foi suspensa no tampão de lise (20 Tris de mm pH8, 100 mM NaCI, 2 mM PMSF) e congelou-se. A pelota de célula então foi descongelada, lisada por ultrassom, e girada em 7,000rcf durante 20 minutos. ID83 é uma proteína de corpo de inclusão. A pelota foi lavada 2x com Sujeitos de 1 %. A pelota foi solubilized em 60 ml_ no tampão obrigatório (8M uréia, 20 pH de Tris de mm 8, 100 mM NaCI) e ligou a 16 ml_ Ni-NTA a resina na TEMPERATURA AMBIENTE durante 1 hora. A resina foi lavada (60 mL 0. δ % DOUTOR durante 20 minutos; 80 IPA de 60 % ml_ durante 30 minutos, 50 ml_ 0.5%DOC enxaguadura) e depois eluído com tampão obrigatório com 300 mM imidazol. O sobrenadante desde o princípio liga esteve ligado a uma 8 resina ml_ adicional e processado como indicado acima. As purificações acima mencionadas removeram produtos de emergência. Outros Ni-NTA ligam foi realizada durante a noite em 4 °C em 160 ml_ (tampão obrigatório com 50 mM NaCI) com 32 resina mL. A resina foi lavada e eluída como indicado acima. As frações disto ligam são dializadas em 20 Tris de mm pH8.
EXEMPLO 18 CLONAGEM E EXPRESSÃO DA PROTEÍNA DE FUSÃO RECOMBINANTE ID94
[00413]Os seguintes iniciadores são usadas em para clonagem a fusão constroem ID94, que compreende os parceiros de fusão derivaram de Rv2389, Rv1813, Rv3620 e Rv2608: 5': Rv2389mat-5Ndel CAATTACATATGGACGACATCGATTGGGACGCC (SEQ ID N°: 93) 3': Rv2389mat-3Kpnl-Hindlll CAATTAAAGCTTTTAAGTACTTGGTTTGCTGCCTCTCGATCG (SEQ ID NO: 94) Rv2389 foi amplificada por PCR do padrão genomic (94 °C para 0,5 minutos, 58 °C para 0,5 minutos, 68 °C para 1.5 minutos, 35 ciclos), digerido com Ndel/Hindlll, e ligam em pET28.a. O id83-pET28.a vetor foi digerido com Kpnl/Hindlll para retirar-se da inserção. ID83 foi ligam em rv2389-pET28.a o vetor (também digerido com Kpnl/Hindlll). O construtor de fusão mandou apresentar uma sequência de polinucleotídeo em SEQ ID NO: 92, codificando a proteína de fusão apresentada na SEQ ID NO: 95. ID94 foi expressado no hospedeiro BL-21 plysS (1 L, 2XYT meio de crescimento, 37 °C). A expressão foi induzida com 1 mM IPTG em OD 0.50 e colhida em OD 1.41. A pelota de célula foi suspensa no tampão de lise (20 Tris de mm pH8, 100 mM NaCI, 2 mM PMSF) e congelou-se. ID94 forma um corpo de inclusão e foi processado o mesmo como ID83. ID94 não ligou bem durante a noite assim o volume foi dobrado com 8M a uréia e BME foram acrescentados a 10 mm. As soluções menos concentradas são ligou a resina Ni-NTA na TEMPERATURA AMBIENTE du- rante 2 horas então durante a noite em 4 °C. A resina foi lavada e eluída como anteriormente indicado. As frações desta purificação são dializadas em 20 Tris de mm pH8.
EXEMPLO 19
CLONAGEM E EXPRESSÃO DA PROTEÍNA DE FUSÃO RECOMBINANTE ID95
[00414]ID95 é um construtor de fusão compreendendo os parceiros de fusão derivaram de Rv2389, Rv3810, Rv1813, Rv3620 e Rv0496. O id58-pET28.a vetor foi digerido com Kpnl/Hindlll para retirar-se da inserção. A inserção de ID58 foi ligam em anteriormente feito 36f. 1 - pET28.a vetor (também digerido com Kpnl/Hindlll). O construtor de fusão mandou apresentar uma sequência de polinucleotídeo em SEQ ID NO: 96, codificando a proteína de fusão apresentada na SEQ ID NO: 97. ID95 foi expressado no hospedeiro BL-21 plysS (1 L, 2XYT meio de crescimento, 37 °C). A pelota de célula foi suspensa no tampão de lise (20 Tris de mm pH8, 100 mM NaCI, 2 mM PMSF) e congelou-se. ID95 forma um corpo de inclusão e foi purificado o mesmo como ID83.
EXEMPLO 20
CLONAGEM E EXPRESSÃO DA PROTEÍNA DE FUSÃO RECOMBINANTE ID120.
[00415]ID120 é um construtor de fusão compreendendo os parceiros de fusão derivaram de Rv2389, Rv3810, Rv1813, Rv3620 e Rv2608. O id83-pET28.a vetor foi digerido com Kpnl/Hindlll para retirar-se da inserção. A inserção de ID83 foi ligam em anteriormente feito 36f. 1 - pET28.a vetor (também digerido com Kpnl/Hindlll). O construtor de fusão mandou apresentar uma sequência de polinucleotídeo em SEQ ID NO: 98, codificando a proteína de fusão apresentada na SEQ ID NO: 99. ID120 foi expressado no hospedeiro BL-21 plysS (1L, 2XYT meio de crescimento, 37 °C). A expressão foi induzida com 1 mM que IPTG em OD 0.50 e células são colhido em OD 1.41. A pelota de célula foi suspensa no tampão de lise (20 Tris de mm pH8, 100 mM NaCI, 2 mM PMSF) e congelou-se. ID120 forma um corpo de inclusão e foi processado o mesmo como ID83. ID120 não ligou bem durante a noite assim o volume foi dobrado com 8M a uréia e BME foram acrescentados a 10 mm. As soluções menos concentradas são ligado à resina Ni-NTA na TEMPERATURA AMBIENTE durante 2 horas então durante a noite em 4 °C. A resina foi lavado e elu- ído como anteriormente indicado. As frações desta purificação são dializadas em 20 Tris de mm pH8.
EXEMPLO 21
RECONHECIMENTO DE ANTÍGENOS MTB POR PPD+ PBMC HUMANO E ESPLENÓCITOS DE MTB CAMUNDONGOS INFECTADOS
[00416]Este exemplo demonstra que o antígeno Mtb de acordo com a invenção induz respostas de revocação de memória em PBMC humano de PPD+ doadores sãos, e esplenócitos isolados de camundongos infectados com Mycobacterium tuberculosis.
MATERIAIS E MÉTODOS:
PBMC humano in vitro estimulação e citoquina ELISA
[00417]PBMC são obtido por apheresis ou purificado do sangue heparinized de 7 PPD-, e 15 PPD+ doadores sãos. PBMC são plaqueados em triplicata 96 - poço placas de cultura de tecido em 2-2.5 x 105 células/poço e cultivado com o meio, PHA (10 pg/mL), Mycobacterium tuberculosis (Mtb) lisado (10 pg/mL), ou cada proteína recombinante (50 pg/mL) por 72 h. Os sobrenadantes são colhido e analisado para IFN-y pelo sanduíche duplo ELISA utilizando de mAb específico (eBioscience), e depois do protocolo do fabricante.
[00418]A Citoquina de Camundongo o Baço de ELISPOT de Mycobacterium tuberculosis - Infecta camundongos é colhido em tempos diferentes pós-infecção, e os suspensões de esplenócito únicos são obtido por procedimentos convencionais. Um ensaio de ELISPOT foi usado para determinar o número relativo de IFN-y ou esplenócitos TNF-expressam. A microplaca de filtração de 96 poços tipo MultiScreen (Millipore, Bedford, Massachusetts) são coberto de 10 pg/mL IFN-y de rato anti- camundongo, ou TNF, capturam Ab (eBioscience) e incubado durante a noite em 4 °C. Placas foram lavados com PBS, bloqueado com 1640 RPMI e FBS de 10 % de pelo menos 1 h em temperatura ambiente, e lavado novamente. As células de baço são plaqueado, em duplicata, em 2 x 105 células/poço, e estimulado com o trapo específico em um 10 pg/mL por 48 h a 37 °C. As placas são posteriormente lavadas com PBS e Tween de 0.1 % e incubado durante a noite em 4 °C com um conjugado pela biotina, anti-camundongo de rato IFN-y, ou TNF, Ab secundário (eBioscience) em 5 pg/mL nPBS, BSA de 0,5 %, e Tween de 0.1 %. Os filtros são o utilizando desenvolvido do ALFA Vectastain avidin peroxidase conjugado e Vectastain AEC kits de substrato (Laboratórios de Vetor, Burlingame, Califórnia) de acordo com o protocolo do fabricante. A reação foi parada lavando as placas com a água de deionizada, as placas são secas no escuro, e as manchas são contadas.
RESULTADOS:
Reconhecimento de proteínas Mtb recombinantes por PPD humano + PBMC,
[00419]PBMC de PPD+ e doadores PPD-foram cultivadas por 72 h com Mtb Rv0164, Rv0455, Rv0496, Rv2608, Rv3020, Rv3478, Rv3619, Rv3620, Rv1738, Rv1813, Rv3810, Rv2389, Rv2866, Rv3876, Rv0054, Rv0410, Rv0655, Rv0831, Rv1009, Rv1099, Rv1240, Rv1288, Rv1410, Rv1569, Rv1789, Rv1818, Rv1860, Rv1886, Rv1908, Rv2220, Rv2032, Rv2623, Rv2875, Rv3044, Rv3310, Rv3881, Rv0577, Rv1626, Rv0733, Rv2520, Rv1253, Rv1980, Rv3628, Rv1884, e proteínas recombinantes Rv1511. Uma descrição da produção destes antígenos recombinantes é descrita em outro lugar aqui. A concentração de IFN-y foi também analisada nos sobrenadantes de cultura de célula.
[00420]Todas as proteínas recombinantes teste, exceto Rv1908, ser presen te a e células T ativadas de PPD+ doadores para produzir IFN-y (Figo 1). Os níveis somente de fundo de IFN-y são detectado em resposta a estes antígenos usando PBMC do controle de PPD-. 5-a aumentos de 70 pregas em IFN-y concentração são medido em culturas PBMC de PPD+ doadores em comparação com o controle de PPD-, indicando antígeno reconhecimento específico destas proteínas recombinantes de doadores anteriormente expostos a Mycobacterium tuberculosis ou Mycobacterium bovis (vacinado com BCG). O reconhecimento de proteínas recombinantes Mtb por esplenócitos de M. tuberculosis - infectando camundongos
[00421]Os camundongos são infectado pela exposição de aerossol de dose baixa com Mycobacterium tuberculosis cepa de H37Rv, e os baços são colhido na pós-infecção de tempo diferente. Um ensaio de ELISPOT foi usado para determinar o número relativo de esplenócitos TNF-expressam em resposta a Rv0164 recombi nante Mtb, Rv0455, Rv0496, Rv2608, Rv3020, Rv3478, Rv3619, Rv3620, Rv1738, Rv1813, Rv3810, Rv2389, Rv2866, Rv0054, Rv0655, Rv0831, Rv1009, Rv1240, Rv1288, Rv1410, Rv1569, Rv1789, Rv1818, Rv1860, Rv1886, Rv1908, Rv2220, Rv2032, Rv2875, Rv3044, Rv3310, Rv3881, Rv0577, Rv1626, Rv0733, Rv1253, Rv1980, Rv3628, Rv1884, Rv3875, Rv1511 e proteínas ID83 durante uns 48 h in vitro cultura.
[00422]Todas as proteínas recombinantes e as proteínas de fusão teste induziu um aumento no número de TNF + os esplenócitos de Mycobacterium tuberculosis - Infectam camundongos s de 28 dias (Figo 2, lista superior), s de 60 dias (dados não mostrados), e pós-infecção s de 90 dias (Figo 2, lista inferior).
[00423]Em kit estes dados indicam que a infecção de Mycobacterium tuber-culosis em camundongos induziu respostas imunes a proteínas Mtb, incluindo em Rv0164, Rv0455, Rv0496, Rv2608, Rv3020, Rv3478, Rv3619, Rv3620, Rv1738, Rv1813, Rv3810, Rv2389, Rv2866, Rv0054, Rv0655, Rv0831, Rv1009, Rv1240, Rv1288, Rv1410, Rv1569, Rv1789, Rv1818, Rv1860, Rv1886, Rv1908, Rv2220, Rv2032, Rv2875, Rv3044, Rv3310, Rv3881, Rv0577, Rv1626, Rv0733, Rv1253, Rv1980, Rv3628, Rv1884, Rv1511 e proteínas ID83.
[00424]Assim, taπto os seres humanos naturalmente expuseram a, e ca-mundongos infectados por um teste de provocação com aerossol com virulento, Mycobacterium tuberculosis - montou respostas imunes a proteínas bacterianas, como evidenciado por respostas de revocação ao lisado Mtb e PPD. Além disso, o aumento em IFN-y e respostas de citoquina TNF a Rv0164, Rv0455, Rv0496, Rv2608, Rv3020, Rv3478, Rv3619, Rv3620, Rv1738, Rv1813, Rv3810, Rv2389, Rv2866, Rv3876, Rv0054, Rv0410, Rv0655, Rv0831, Rv1009, Rv1099, Rv1240, Rv1288, Rv1410, Rv1569, Rv1789, Rv1818, Rv1860, Rv1886, Rv1908, Rv2220, Rv2032, Rv2623, Rv2875, Rv3044, Rv3310, Rv3881, Rv0577, Rv1626, Rv0733, Rv2520, Rv1253, Rv1980, Rv3628, Rv1884, Rv1511 e proteína ID83 sobre in vitro a estimulação indica que estes antígenos (1) são reconhecidos por anteriormente os indivíduos expostos (a presença da memória T células), (2) podem ser usados como a immuno-terapêutica ou (3) pode ser usada como diagnóstica.
EXEMPLO 22 RESPOSTAS IMUNES A ANTÍGENOS MTB EM CAMUNDONGOS C57BL/6 E PROTEÇÃO CONTRA DESAFIO DE AEROSSOL COM MTB
[00425]Este exemplo demonstra que a imunização de camundongos com antígenos Mtb de acordo com a invenção é imunogênica e pode fornecer a proteção contra o aerossol desafio de Mycobacterium tuberculosis.
MATERIAL & MÉTODOS:
Antígenos recombinantes e formulações de adjuvante
[00426]As proteínas recombinantes são produzido tal como descrito acima. CpG 1826 foi obtido de Produtos farmacêuticos Coley (Wellesley, Massachusetts).
[00427]A Imunização camundongos C57/BL6 Femininos é obtido de Charles River e idade - combinado (5-7 semana) dentro de cada experimento. Os camundongos são imunizado três vezes (de 3 semanas à parte) com 8 pg de Rv0164 recombinante (SEQ ID NO: 1), Rv0496 (SEQ ID NO: 6), Rv2608 (SEQ ID NO: 26), Rv3020 (SEQ ID NO: 36), Rv3478 (SEQ ID NO: 41), Rv3619 (SEQ ID NO: 46), Rv3620 (SEQ ID NO: 51), Rv1738 (SEQ ID NO: 11), Rv1813 (SEQ ID NO: 16), Rv3810 (SEQ ID NO: 56), Rv2389 (SEQ ID NO: 21), Rv2866 (SEQ ID NO: 31), Rv0831 (SEQ ID NO: 115), Rv1288 (SEQ ID NO: 127), Rv1569 (SEQ ID NO: 133), Rv1789 (SEQ ID NO: 136), Rv1818 (SEQ ID NO: 139), Rv1860 (SEQ ID NO: 142), Rv1886 (SEQ ID NO: 145), Rv2220 (SEQ ID NO: 154), Rv2032 (SEQ ID NO: 151), Rv2623 (SEQ ID NO: 160), Rv2875 (SEQ ID NO: 163), Rv3044 (SEQ ID NO: 166), Rv0577 (SEQ ID NO: 184), Rv1626 (SEQ ID NO: 187), Rv0733 (SEQ ID NO: 190), Rv3628 (SEQ ID NO: 202), e Rv1884 (SEQ ID NO: 205) proteína formulada com 25 pg do adjuvante CpG. Os camundongos na salina, adjuvante somente, e grupos de controle da BCG receberam três doses dPBS, três doses do adjuvante sozinho, ou a dose única de 5 x 104 BCG CFU respectivamente. Os camundongos são injetado com um volume total de 100pl/mouse via a via s.c..
Citoquina ELISA
[00428]Três semanas depois do último estímulo, o baço de animais nomeados para estudos de imunogenicidade é colhido, e os esplenócitos são obtido por procedimentos convencionais. Para a análise de citoquina, os esplenócitos são pla- queados em duplicata 96 - poço placas de cultura de tecido em 2.5 x 105 célu- las/poço e cultivado com o meio, Con A 3 pg/mL, PPD 10 pg/mL, lisado de Mtb 10 pg/mL, ou cada proteína recombinante 10 pg/mL de 72 h. Os sobrenadantes são colhido e analisado para IFN-y pelo sanduíche duplo ELISA utilizando de mAb específico (eBioscience), e depois do protocolo do fabricante.
Citoquina ELISPOT.
[00429]As microplacas de filtração de 96 poços tipo MultiScreen (Millipore, Bedford, Massachusetts) são cobertas de 10 pg/mL IFN-y de rato anti-camuπdongo ou TNF capturam Ab (eBioscience) e incubado durante a noite em 4 °C. Placas foram lavados com PBS, bloqueado com 1640 RPM I e FBS de 10 % de pelo menos 1 h em temperatura ambiente, e lavado novamente. Os esplenócitos são plaqueados em duplicata em 2 x 105 células/poço, e estimulado com o meio, Con A 3 pg/mL, PPD 10 pg/mL, ou cada proteína recombinante 10 pg/mL por 48 h a 37 °C. As placas são posteriormente lavadas com PBS e 0,1 % de Tween-20 e incubado por 2 h com um anti-camundongo de rato conjugado pela biotina IFN-y ou Ab secundário TNF (eBioscience) em 5 pg/mL nPBS, BSA de 0,5 %, e 0,1 % de Tween-20. Os filtros são o utilizando desenvolvido do ALFA Vectastain avidin peroxidase conjugado e Vectastain AEC kits de substrato (Laboratórios de Vetor, Burlingame, Califórnia) de acordo com o protocolo do fabricante. A reação foi parada lavando as placas com a água de deionizada, as placas são secas no escuro, e as manchas são contado sobre uma automatizou o leitor ELISPOT (C.T.L. Serie3A Analisador, Cellular Technology Ltd, Cleveland, Ohio), e analisado com Immunospot ® (CTL Analisador LLC).
ELISA de isotipo de IgG.
[00430]Os animais são sangrou 1 wk depois da imunização última e soro que lgG1 e as titulações de anticorpo lgG2c são determinado. As placas de Polisor- veira de Nunc-lmmuno são coberto para 4 h na temperatura ambiente com 2 pg/mL da proteína recombinante no tampão de bicarbonato de 0.1 m, bloqueado durante a noite em 4 °C com PBS Tween-20 a 0,05 % e BSA a 1 %, lavado com PBS Tween- 20 0,05 %, incubados por 2 h na temperatura ambiente com soros em um 1:50 diluição e diluições seriais de 5 pregas subsequentes, lavadas, e incubado para 1 h com anti-lgG1-HRP ou anti-lgG2c-HRP 1:2000 nPBS Tween-20 BSA de 0,05 % 0.1 %. Placas foram lavados e utilizando desenvolvido SureBlue TMB substrato (KPL Inc, Gaithersburg, Maryland). A reação enzimática foi parada com 1 N H2SO4, e as pla- cas são lido dentro de 30 min em 450 nm com urn kit de filtro de referência em 650 nm utilizando de uma microplaca leitor de ELISA (Dispositivos Moleculares, Sunnyvale, Califórnia) e SoftMax Pro5. As titulações de ponto final são determinado com o Prisma GraphPad 4 (GraphPad Software Inc. São Diego, Califórnia) com um atalho de 0.1.
Experimento de proteção
[00431]Os camundongos são s.c imunizado, três vezes, 3 semanas à parte, com 8 Dg de cada proteína recombinante de um subkit de antígenos Mtb, e mistura com o adjuvante CpG. Os camundongos de controle seguros são imunizado com BCG (5 x 104 CFU) na base da cauda (uma vez), e os animais de controle negativos são injetado com a salina, ou adjuvante sozinho. S de trinta dias depois da imunização última, os camundongos são provocado pela exposição de aerossol de dose baixa com Mycobacterium tuberculosis cepa de H37Rv (ATCC 35718; Coleção de Cultura de Tipo americana, Manassas, VA) utilizando de uma câmara de exposição de aerossol de UW-MADISON (Madison, Wl) calibrado para liberar 50-100 bactérias nos pulmões. Quatro semanas depois, os camundongos são euthanized, e o pulmão e o baço homogenates foram preparados em PBS/Tween 80 (0.05 %). As contas bacterianas são determinam pela galvanização diluições seriais de órgãos inteiros individuais em Middlebrook nutriente 7H11 Ágar-ágar de Bacto (BD Biosciences, Cockeysville, Maryland) e contagem de formação de colônia bacteriana depois da incubação de 14 dias em 37 °C no ar umedecido e CO2 de 5 %. Os dados são expressados como Log10 do número avaro de bactérias recuperou SD ±, e Redução Log10 de CFU = Log 10 CFU do grupo vacinado - Log 10 CFU do grupo tratado Salino.
RESULTADOS:
[00432]Respostas imunes a antígenos Mtb recombinantes adjuvanted com CpG. Os camundongos de C57BL/6 são imunizado três vezes, três semanas à parte, com Mtb recombinante Rv0164, Rv0455, Rv0496, Rv2608, Rv3020, Rv3478, Rv3619, Rv3620, Rv1738, Rv1813, Rv3810, Rv2389, Rv2866, Rv0831, Rv1818, Rv1886, Rv2032, Rv2623, Rv2875, Rv3044, Rv0577, Rv1626, Rv3628, e proteínas Rv1884 formuladas com 25 pg do adjuvante CpG. Uma semana, e três semanas depois da imunização última, a presença do antígeno anticorpo específico, e memória T linfócitos respectivamente, é avaliado.
[00433]O soro específico IgG isotipo Ab resposta foi medido por ELISA con-vencional pelo revestimento cada uma da proteína recombinante sobre uma placa e em série diluição dos soros diferentes, as proporções de titulação de ponto final de lgG2c:lgG1 são determinado para cada grupo de vacina (a Tabela 1). A salina, adju-vante de CpG sozinho, ou imunização da BCG não induziu um lgG1 ou resposta de anticorpo lgG2c específica para qualquer ou as proteínas recombinantes Mtb teste (dados não mostrados). A imunização com cada uma das proteínas recombinantes Mtb com o adjuvante CpG induziu o antígeno lgG1 específico e lgG2c. A tabela 1. Respostas imunes a antígenos Mtb
[00434]auma unidade da formação da Mancha por milhão de células (SD). Os camundongos são s.c. imunizado três vezes, três semanas à parte com antígenos Mtb (Rv#) + CpG. As respostas de citoquina aos antígenos são determinado por ELISPOT 3 semanas depois da injeção última. b lgG2c:lgG1 proporção, *P <0.05, o Teste de t de Estudante, ° ND, não feito.
[00435]Três semanas depois da imunização última, os esplenócitos foram preparados e provaram por ELISPOT determinar o número relativo de IFN-y ou es-plenócitos TNF-expressam em resposta ao meio sozinho, o mitogen ConA, PPD, lisado de Mtb, e cada uma das proteínas Mtb recombinantes.
[00436]A injeção com a salina, ou adjuvante CpG sozinho não induziu IFN-y ou respostas TNF específicas para qualquer das proteínas recombinantes (dados não mostrados).
[00437]A imunização com cada uma das proteínas recombinantes Mtb com o adjuvante CpG induziu o antígeno IFN-específico y e/ou respostas de revocação de TNF por esplenócitos ativados (a Tabela 1). Os níveis inferiores de IFN-y em resposta ao lisado Mtb e PPD são também observado (dados não mostrados), sugerindo que estas proteínas sejam naturalmente encontradas em lisados de micobactéria e derivados parcialmente purificados.
[00438]Em kit, estes resultados indicam que a imunização com os antígenos Mtb recombinantes diferentes em CpG induziu uma resposta de memória do tipo Th1 com lgG2c predominante, IFN-y, eTNF.
[00439]A proteção permitida pelas proteínas recombinantes Mtb diferentes, adjuvanted com CpG, contra um aerossol desafia com Mtb H37Rv. O número de bacilos viáveis, expressados como média Log10 CFU, no pulmão e o baço de camundongos vacinados com a proteína recombinante Mtb que Rv0496, Rv2608, Rv3020, Rv3478, Rv3619, Rv3620, Rv1813, Rv1569, Rv1789, Rv1860, Rv1886, Rv2220, Rv2875, Rv3044, Rv0577, Rv1626, e Rv0733, adjuvanted com CpG, são determinado 4 semanas põe o desafio de aerossol no correio com ~50 CFU de Mycobacterium tuberculosis virulento H37Rv. O média Log10 CFU no pulmão de camundongos imunizados com as proteínas recombinantes diferentes foi em compa- ração com o média Log10 CFU obtido em camundongos que recebem placebo (salina) ou BCG, o atual e somente vacina contra TB. A diferença no Log avaro 10 CFU no grupo salino contra os grupos vacinados é expressada como Log 10 redução de CFU.
[00440]A imunização de camundongos com três doses de Rv3478 + CpG ou Rv2608 + CpG resultou em uma redução em bacilos Mtb viáveis, no pulmão (0.66, respectivamente 0.58) perto disto permitido pela vacinação da BCG (0.78) (a Tabela 2). A imunização com cada um de Rv0496, Rv3020, Rv3619, Rv3620, Rv1813, Rv1569, Rv1789, Rv1860, Rv1886, Rv2220, Rv2875, Rv3044, Rv0577, Rv1626, e Rv0733, adjuvanted com CpG, também permitiu a proteção pouca contra a infecção Mtb. O adjuvante de CpG sozinho não reduziu o pulmão carga bacteriana (-0.09). A tabela 2. Proteção induzida pela vacina contra Mtba
[00441]Camundongos são s.c. imunizado três vezes, três semanas à parte com 8 pg Mtb antígenos (Rv#) + 25 pg CpG. b
[00442]bRedução de bactérias viáveis (CFU) nos pulmões em comparação com a salina imunizaram animais 4 semanas depois de um teste de provocação com aerossol de dose baixa com M. tuberculosis cepa de Erdman ou H37Rv.
[00443]Estes resultados são surpresa nisto os níveis da proteção contra a in-fecção Mtb são atingido com 3 doses de uma proteína recombinante única adjuvanted com CpG.
EXEMPLO 23
RESPOSTAS IMUNES A UMA MISTURA DE ANTÍGENOS MTB EM CAMUNDONGOS C57BL/6 E PROTEÇÃO CONTRA DESAFIO DE AEROSSOL COM MTB
[00444]Este exemplo demonstra que a imunização de camundongos com uma mistura de antígenos Mtb de acordo com a invenção é imunogênica e pode fornecer a proteção contra o aerossol desafio de Mycobacterium tuberculosis.
MATERIAL & MÉTODOS:
Antígenos recombinantes e formulações de adjuvante
[00445]As proteínas recombinantes são produzido tal como descrito acima. CpG 1826 foi obtido de Produtos farmacêuticos Coley (Wellesley, Massachusetts).
Imunização
[00446]Os camundongos C57/BL6 femininos são obtido de Charles River e idade - combinado (5-7 semana) dentro de cada experimento. Os camundongos são imunizado três vezes (de 3 semanas à parte) com 6 ou 8 pg de Rv2608 recombinante, Rv3620, e proteína Rv1813 formulada com 25 pg do adjuvante CpG. Os camundongos no adjuvante somente, e os grupos de controle da BCG receberam três doses do adjuvante sozinho, ou uma dose única de 5 x 104 BCG CFU respectivamente. Os camundongos são injetado com um volume total de 10Opl/mouse via a via s.c..
Citoquina ELISA
[00447]Três semanas depois do último estímulo, o baço de animais nomeados para estudos de imunogenicidade é colhido, e os esplenócitos são obtido por procedimentos convencionais. Para a análise de citoquina, os esplenócitos são pla- queados em duplicata 96 - poço placas de cultura de tecido em 2.5 x 105 célu- las/poço e cultivado com o meio, Con A 3 pg/mL, PPD 10 pg/mL, lisado de Mtb 10 pg/mL, ou cada proteína recombinante 10 pg/mL de 72 h. Supernatants são colhido e analisado para IFN-y pelo sanduíche duplo ELISA utilizando de mAb específico (eBioscience), e depois do protocolo do fabricante. Citoquina a Multitela 96 de ELISPOT - poço as placas de filtração (Millipore, Bedford, Massachusetts) são co- berto de 10 pg/mL IFN-y de rato anti-camuπdongo ou TNF capturam Ab (eBioscience) e incubado durante a noite em 4 °C. Placas foram lavados com PBS, bloqueado com 1640 RPML e FBS de 10 % de pelo menos 1 h em temperatura ambiente, e lavado novamente. Os esplenócitos são plaqueados em duplicata em 2 x 105 célu- las/poço, e estimulado com o meio, Con A 3 pg/mL, PPD 10 pg/mL, ou cada proteína recombinante 10 pg/mL por 48 h a 37 °C. As placas são posteriormente lavadas com PBS e 0,1 % de Tween-20 e incubado por 2 h com um anti-camundongo de rato conjugado pela biotina IFN-y ou Ab secundário TNF (eBioscience) em 5 pg/mL nPBS, BSA de 0,5 %, e 0,1 % de Tween-20. Os filtros são o utilizando desenvolvido do ALFA Vectastain avidin peroxidase conjugado e Vectastain AEC kits de substrato (Laboratórios de Vetor, Burlingame, Califórnia) de acordo com o protocolo do fabricante. A reação foi parada lavando as placas com a água de deionizada, as placas são secas no escuro, e as manchas são contado sobre uma automatizou o leitor ELISPOT (C.T.L. Serie3A Analisador, Cellular Technology Ltd, Cleveland, Ohio), e analisado com Immunospot ® (CTL Analisador LLC).
ELISA de isotipo de IgG
[00448]Os animais são sangrou 1 wk depois da imunização última e soro que lgG1 e as titulações de anticorpo lgG2c são determinado. As placas de Polisor- veira de Nunc-lmmuno são coberto para 4 h na temperatura ambiente com 2 pg/mL da proteína recombinante no tampão de bicarbonato de 0.1 m, bloqueado durante a noite em 4 °C com PBS Tween-20 a 0,05 % e BSA a 1 %, lavado com PBS Tween- 20 0,05 %, incubados por 2 h na temperatura ambiente com soros em um 1:50 diluição e diluições seriais de 5 pregas subsequentes, lavadas, e incubado para 1 h com anti-lgG1-HRP ou anti-lgG2c-HRP 1:2000 nPBS Tween-20 BSA de 0,05 % 0.1 %. Placas foram lavados e utilizando desenvolvido SureBlue TMB substrato (KPL Inc, Gaithersburg, Maryland). A reação enzimática foi parada com 1 N H2SO4, e as placas são lido dentro de 30 min em 450 nm com um kit de filtro de referência em 650 nm utilizando de uma microplaca leitor de ELISA (Dispositivos Moleculares, Sunnyvale, Califórnia) e SoftMax Pro5. As titulações de ponto final são determinado com o Prisma GraphPad 4 (GraphPad Software Inc. São Diego, Califórnia) com um atalho de 0.1.
Experimento de proteção
[00449]Os camundongos são s.c imunizado, três vezes, 3 semanas à parte, com 6 ou 8 pg de cada proteína recombinante de um subkit de antígenos Mtb, e mistura com o adjuvante CpG. Os camundongos de controle seguros são imunizado com BCG (5 x 104 CFU) na base da cauda (uma vez), e os animais de controle negativos são injetado com o adjuvante sozinho. S de trinta dias depois da imunização última, os camundongos são provocado pela exposição de aerossol de dose baixa com Mycobacterium tuberculosis cepa de H37Rv (ATCC 35718; Coleção de Cultura de Tipo americana, Manassas, VA) utilizando de uma câmara de exposição de aerossol de UW-MADISON (Madison, Wl) calibrado para liberar 50-100 bactérias nos pulmões. Quatro semanas depois, os camundongos são euthanized, e o pulmão e o baço homogenates foram preparados em PBS/Tween 80 (0.05 %). As contas bacte- rianas são determinam pela galvanização diluições seriais de órgãos inteiros individuais em Middlebrook nutriente 7H11 Ágar-ágar de Bacto (BD Biosciences, Cockeysville, Maryland) e contagem de formação de colônia bacteriana depois da incubação de 14 dias em 37 °C no ar umedecido e CO2 de 5 %. Os dados são expressados como Log10 do número avaro de bactérias recuperou SD ±, e Redução Log10 de CFU = Log10 CFU para o grupo vacinado - Log10 CFU para o grupo tratado Salino.
RESULTADOS: respostas imunes a uma mistura de antígenos Mtb recombi-nantes adjuvanted com CpG.
[00450]Os camundongos de C57BL/6 são imunizado três vezes, três semanas à parte, com cada Mtb recombinante Rv2608, Rv3620, e proteínas Rv1813, se- paradamente (8pg) ou em uma mistura (6 pg cada um), formulado com 25 pg do ad-juvante CpG. Uma semana, e três semanas depois da imunização última, a presença do antígeno anticorpo específico, e memória T linfócitos respectivamente, é avaliado.
[00451]O soro específico IgG isotipo Ab resposta foi medido por ELISA con-vencional pelo revestimento cada uma da proteína recombinante sobre uma placa e em série diluição dos soros diferentes, as titulações de ponto final de lgG2c são de-terminado para cada grupo de vacina. O adjuvante de CpG sozinho ou imunização da BCG não induziu um lgG1 ou resposta de anticorpo lgG2c específica para qualquer ou as proteínas recombinantes Mtb teste (a Figura 3B, e dados não mostrados). A imunização com cada uma das proteínas recombinantes Mtb com o adjuvante CpG induziu o antígeno lgG1 específico (dados não mostrados) e lgG2c (a Figura 3B).
[00452]Três semanas depois da imunização última, os esplenócitos foram preparados e provaram por ELISA ou ELISPOT determinar o nível relativo de IFN-y ou número de esplenócitos TNF-expressam em resposta ao meio sozinho, o mitogen ConA, PPD, lisado de Mtb, e cada uma das proteínas Mtb recombinantes.
[00453]A injeção com o adjuvante CpG sozinho não induziu IFN-y ou respostas TNF específicas para qualquer das proteínas recombinantes (a Figura 3C-D).
[00454]A imunização com cada uma das proteínas recombinantes Mtb com o adjuvante CpG induziu o antígeno IFN-específico y e respostas de revocação de TNF por esplenócitos ativados (a Figura 3C-D). Os níveis inferiores de respostas de citoquina são observado quando os três antígenos são usadas como uma mistura.
[00455]Em kit, estes resultados indicam que a imunização com os antígenos Mtb recombinantes diferentes, separadamente ou como uma mistura, em CpG induziu uma resposta de memória do tipo Th1 com lgG2c predominante, IFN-y, e TNF.
[00456]A proteção permitida por uma mistura de proteínas recombinantes Mtb diferentes, adjuvanted com CpG, contra um aerossol desafia com Mtb H37Rv.
[00457]0 número de bacilos viáveis, expressados como média Log10 CFU, no pulmão de camundongos vacinados com a proteína recombinante Mtb que Rv2608, Rv3620, e Rv1813, separadamente (8pg) ou em uma mistura (6 pg cada um), adjuvanted com CpG, são determinado 4 semanas põe o desafio de aerossol no correio com-50 CFU de Mycobacterium tuberculosis virulento H37Rv. O média Log10 CFU no pulmão de camundongos imunizados com as proteínas recombinantes diferentes foi em comparação com o média Log10 CFU obtido em camundongos que recebem adjuvante sozinho ou BCG, o atual e somente vacina contra TB. A diferença no média Log10 CFU no grupo de adjuvante contra os grupos vacinados é expressada como redução de Log10 de CFU.
[00458]A imunização de camundongos com três doses de Rv2608 + Rv3620 + Rv1813 + CpG resultou em uma redução em bacilos Mtb viáveis no pulmão (redução de Log10 de CFU de 0.67) perto disto permitido pela vacinação da BCG (0.71) (a Figura 3A). A imunização com Rv2608 ou Rv1813, adjuvanted com CpG, também permitiu a proteção pouca contra a infecção Mtb (0.24 e 0.30 respectivamente). A imunização com Rv3620 + CpG ou adjuvante CpG sozinho não reduziu o pulmão carga bacteriana. A redução de CFU atingido injetando uma mistura de três antígenos Mtb foi mais alta do que a soma de efeitos individuais. Estes resultados são surpresa nisto os níveis da proteção contra a infecção Mtb são aumentado com 3 doses de uma mistura ou três proteínas recombinantes adjuvanted com CpG, em comparação com 3 doses de proteínas individuais com CpG.
O EXEMPLO 24 RESPOSTAS IMUNES a ID83 E PROTEÍNAS DE FUSÃO ID93 em CAMUNDONGOS C57BL/6 E PROTEÇÃO CONTRA O DESAFIO DE AEROSSOL com MTB
[00459]Este exemplo demonstra que a imunização de camundongos com proteínas de fusão de acordo com a invenção é imunogênica e pode fornecer a pro- teção contra o aerossol desafio de Mycobacterium tuberculosis.
MATERIAL & MÉTODOS:
Proteínas de fusão e formulações de adjuvante
[00460]As proteínas de fusão são produzido tal como descrito acima. CpG 1826 foi obtido de Produtos farmacêuticos Coley (Wellesley, Massachusetts). O lipídio A de Glucopyranosyl (GLA) foi obtido de Avanti (Alabastro, a Alabama) e Gardi- quimod (o GDQ) foi obtido de Invivogen (São Diego, Califórnia). As emulsões de zibelina de óleo na água (-SE) foram preparadas por técnicas padrão.
Imunização
[00461]Os camundongos C57/BL6 femininos são obtido de Charles River e idade - combinado (5-7 semana) dentro de cada experimento. Os camundongos são imunizado três vezes (de 3 semanas à parte) com 8 pg de ID83 e proteína de fusão ID93 formulada com 20 pg do GLA-SE de adjuvante, ou 8 pg ID83 proteína de fusão formulada com 20-25 pg do GLA-SE de adjuvante, GDQ-SE, CpG-SE, GLA/GDQ- SE, GLA/CpG-SE, CpG/GDQ-SE. Os camundongos na salina, adjuvante somente, e grupos de controle da BCG receberam três doses dPBS, três doses do adjuvante sozinho, ou uma dose única de 5 x 104 BCG CFU respectivamente. Os camundongos são injetado com um volume total de 100pl/mouse via a via s.c..
Citoquina ELISA
[00462]Três semanas depois do último estímulo, o baço de animais nomeados para estudos de imunogenicidade é colhido, e os esplenócitos são obtido por procedimentos convencionais. Para a análise de citoquina, os esplenócitos são pla- queados em duplicata 96 - poço placas de cultura de tecido em 2.5 x 105 célu- las/poço e cultivado com o meio, Con A 3 pg/mL, PPD 10 pg/mL, lisado de Mtb 10 pg/mL, ou cada proteína de fusão 10 pg/mL de 72 h. Os sobrenadantes são colhido e analisado para IFN-y pelo sanduíche duplo ELISA utilizando de mAb específico (eBioscience), e depois do protocolo do fabricante.
ELISA de isotipo de IgG
[00463]Os animais são sangrou 1 wk depois da imunização última e soro que lgG1 e as titulações de anticorpo lgG2c são determinado. As placas de Polisor- veira de Nunc-lmmuno são coberto para 4 h na temperatura ambiente com 2 pg/mL da proteína recombinante no tampão de bicarbonato de 0.1 m, bloqueado durante a noite em 4 °C com PBS Tween-20 a 0,05 % e BSA a 1 %, lavado com PBS Tween- 20 0,05 %, incubados por 2 h na temperatura ambiente com soros em um 1:50 diluição e diluições seriais de 5 pregas subsequentes, lavadas, e incubado para 1 h com anti-lgG1-HRP ou anti-lgG2c-HRP 1:2000 nPBS Tween-20 BSA de 0,05 % 0.1 %. Placas foram lavados e utilizando desenvolvido SureBlue TMB substrato (KPL Inc, Gaithersburg, Maryland). A reação enzimática foi parada com 1 N H2SO4, e as placas são lido dentro de 30 min em 450 nm com um kit de filtro de referência em 650 nm utilizando de uma microplaca leitor de ELISA (Dispositivos Moleculares, Sunnyvale, Califórnia) e SoftMax Pro5. As titulações de ponto final são determinado com o Prisma GraphPad 4 (GraphPad Software Inc. São Diego, Califórnia) com um atalho de 0.1.
Experimento de proteção
[00464]Os camundongos são s.c imunizado, três vezes, 3 semanas à parte, com 8 pg da proteína de fusão, formulada no adjuvante indicado. Os camundongos de controle seguros são imunizado com BCG (5 x 104 CFU) na base da cauda (uma vez), e os animais de controle negativos são injetado com a salina, ou adjuvante sozinho. S de trinta dias depois da imunização última, os camundongos são provocado pela exposição de aerossol de dose baixa com Mycobacterium tuberculosis cepa de H37Rv (ATCC 35718; Coleção de Cultura de Tipo americana, Manassas, VA) utilizando de uma câmara de exposição de aerossol de UW-MADISON (Madison, Wl) calibrado para liberar 50-100 bactérias nos pulmões. Quatro semanas depois, os camundongos são euthanized, e o pulmão e 0 baço homogenates foram preparados em PBS/Tween 80 (0.05 %). As contas bacterianas são determinam pela galvanização diluições seriais de órgãos inteiros individuais em Middlebrook nutriente 7H11 Ágar-ágar de Bacto (BD Biosciences, Cockeysville, Maryland) e contagem de formação de colônia bacteriana depois da incubação de 14 dias em 37 °C no ar umedeci- do e CO2 de 5 %. Os dados são expressados como Log10 do número avaro de bactérias recuperou ± SDi e Redução Log10 de CFU = Log10 CFU para o grupo vacinado - Log10 CFU para o grupo tratado Salino.
RESULTADOS:
Respostas Imunes a ID83 e ID93 adjuvanted com GLA-SE
[00465]Os camundongos de C57BL/6 são imunizado três vezes, três semanas à parte, com ID83 ou proteínas de fusão ID93 formuladas com 20 pg do GLA-SE de adjuvante. Uma semana, e três semanas depois da imunização última, a presença do antígeno anticorpo específico, e memória T linfócitos respectivamente, é avaliado.
[00466]0 soro específico IgG isotipo Ab resposta foi medido por ELISA con-vencional pelo revestimento cada uma da proteína recombinante sobre uma placa e em série diluição dos soros diferentes. As titulações de ponto final são determinado para cada grupo de vacina. A salina não induziu um lgG1 ou resposta de anticorpo lgG2c específica para ID83 ou proteínas de fusão ID93 (a Figura 4A) nem ENVIDRAÇOU o adjuvante sozinho (dados não mostrados). A imunização com ID83 ou a proteína de fusão ID93 com o adjuvante ENVIDRAÇAM o antígeno induzido lgG1 específico e lgG2c.
[00467]Três semanas depois da imunização última, os esplenócitos foram preparados e provaram por ELISA determinar o nível relativo de IFN-y produzido por esplenócitos em resposta ao meio sozinho, o mitogen ConA, e cada uma das proteínas de fusão. A injeção com salina ou adjuvante de GLA-SE sozinho não induziu respostas IFN-y específicas para ID83 ou proteínas de fusão ID93 (dados não mos- trados).
[00468]A imunização com ID83 ou a proteína de fusão ID93 com o adjuvante ENVIDRAÇAM o antígeno induzido IFN-específico y respostas de revocação por esplenócitos ativados (a Figura 4B).
[00469]Em kit, estes resultados indicam que a imunização com as proteínas de fusão diferentes no GLA-SE induziu B e célula T respostas imunes, o Immunogenicity de ID83 formulado com adjuvantes diferentes
[00470]Os camundongos de C57BL/6 são imunizado três vezes, três semanas à parte, com a proteína de fusão ID83 formulada com 20-25 pg do GLA-SE de adjuvante, GDQ-SE, CpG-SE, GLA/GDQ-SE, GLA/CpG-SE, CpG/GDQ-SE. Uma semana, e três semanas depois da imunização última, a presença do antígeno anticorpo específico, e memória T linfócitos respectivamente, é avaliado.
[00471]O soro específico IgG isotipo Ab resposta foi medido por ELISA con-vencional pelo revestimento cada uma da proteína recombinante sobre uma placa e em série diluição dos soros diferentes. As titulações de ponto final são determinado para cada grupo de vacina. A salina não induziu um lgG1 ou resposta de anticorpo lgG2c específica para proteínas de fusão ID83. A imunização com ID83 com os adjuvantes diferentes induziu o antígeno lgG1 específico e lgG2c (a Figura 5A). Três semanas depois da imunização última, os esplenócitos foram preparados e provaram por ELISA determinar o nível relativo de IFN-y produzido por esplenócitos em resposta ao meio sozinho, o mitogen ConA, e proteína de fusão ID83.
[00472]A injeção com a salina não induziu IFN-y respostas específicas para a proteína de fusão ID83. A imunização com a proteína de fusão ID83 com os adjuvantes diferentes induziu o antígeno IFN-específico v respostas de revocação por esplenócitos ativados (a Figura 5B).
[00473]Em kit, estes resultados indicam que a imunização com a proteína de fusão ID83 em vários adjuvantes induziu B e célula T respostas imunes. A proteção permitida por ID83 e proteínas de fusão ID93, formuladas com o GLA-SE de adjuvante, contra um aerossol desafia com Mtb H37Rv.
[00474]O número de bacilos viáveis, expressados como média Log10 CFU, no pulmão de camundongos vacinados com ID83 ou proteínas de fusão ID93 adjuvanted com o GLA-SE, é determinado 4 semanas põem o desafio de aerossol no correio com-50 CFU de M. tuberculosis virulento H37RV.
[00475]O média LoglO CFU no pulmão de camundongos imunizados com as proteínas de fusão diferentes foi em comparação com o média LoglO CFU obtido em camundongos que recebem placebo (salina) ou BCG. A diferença no média LoglO CFU no grupo salino contra os grupos vacinados é expressada como redução de LoglO de CFU.
[00476]A imunização de camundongos com três doses de ID83 + GLA-SE ou ID93 + GLA-SE resultou em uma redução em bacilos Mtb viáveis no pulmão de camundongos Mtb-infectados de 0.34, respectivamente 0.48 LoglO (a Tabela 3). Estes resultados demonstram que a proteção contra a infecção Mtb foi atingida com 3 doses de duas proteínas de fusão diferentes adjuvanted com o GLA-SE. A tabela 3. Número de bacilos viáveis no pulmão de camundongos vacinados.
[00477]CFU = unidades da formação da colônia. Os valores representam o número de bacilos viáveis nos pulmões de camundongos infectados e são expressados como Logw.
[00478]bDifference = LoglO CFU para o grupo Salino - IO do Log CFU do grupo tratado vacinado. CN/A = não aplicável.
[00479]A proteção permitida por ID83 formulado com adjuvantes diferentes, em camundongos C57BU6, contra um aerossol desafia com Mtb H 37 Rv. O número de bacilos viáveis, expressados como média LoglO CFU, no pulmão de camundongos vacinados com a proteína de fusão ID83 formulada com 20-25 pg do GLA-SE de adjuvante, CpG-SE, ou GLA/CpG-SE é determinado 4 semanas põem o desafio de aerossol no correio com ~50 CFU de M. tuberculosis virulento H37Rv.
[00480]O média LoglO CFU no pulmão de camundongos imunizados com ID83 nos adjuvantes diferentes foi em comparação com o média LoglO CFU obtido em camundongos que recebem placebo (salina) ou BCG. A diferença no média LoglO CFU no grupo salino contra os grupos vacinados é expressada como redução de LoglO de CFU.
[00481]A imunização de camundongos com três doses de ID83 com adju-vantes diferentes resultou em uma redução em bacilos Mtb viáveis no pulmão de camundongos Mtb-infectados (a Tabela 4). Estes resultados são promessa naquela proteção contra a infecção Mtb foi atingido com 3 fins de duas proteínas de fusão diferentes adjuvanted com o GLA-SE.A tabela 4. Número de bacilos viáveis no pulmão de camundongos vacinados.
[00482]CFU = unidades da formação da colônia. Os valores representam o número de bacilos viáveis nos pulmões de camundongos infectados e ser expressados como Logio.
bSD, desvio padrão
[00483]Redução de CCFU = Logw CFU para o grupo Salino - Logw CFU pa- ra o grupo tratado vacinado. o valor de Pd é calculado com ANOVA descartável seguido por múltiplo Teste de comparação de Dunnett. P valores <0.05 são considerados estatisticamente significativo
[00484]Em kit, estes resultados indicam que a vacinação com a proteína de fusão ID83 adjuvanted com CpG-SE, GLA-SE, ou CpG/GLA-SE reduziu a carga bac- teriana e parcialmente protegeu camundongos da infecção por M. tuberculosis. ID83 + CpG/GLA-SE foi a formulação bem mais eficaz em reduzir o número de bactérias viáveis nos pulmões de camundongos Mtb-infectados.
[00485]A proteção permitida por ID83 formulado com GLA/CpG-SE, em por- cos-da-índia, contra um aerossol desafia com Mtb H 37 Rv. A sobrevivência de por- cos-da-índia vacinados com a proteína de fusão ID83 formulada com 20/25 pg do adjuvante GLA/CpG-SE é seguido para o desafio de aerossol de correio por 200 dias com-50 CFU de M. tuberculosis virulento H37Rv.
[00486]A sobrevivência de porcos-da-índia imunizados com ID83 no adjuvante GLA/CpG-SE foi em comparação com a sobrevivência de porcos-da-índia que recebem placebo (salina) ou BCG.
[00487]A imunização de porcos-da-índia com três doses de ID83 com adju-vantes diferentes resultou na sobrevivência aumentada de Mtb - infectam o porco de Guiné (a Figura 6). No dia 200 pós-infecção, 75 % dos animais vacinados com ID83 + GLA/CpG-SE são ainda vivo, comparado com 25 % dos porcos-da-índia no grupo de placebo. 62 % de porcos-da-índia imunizados com BCG são vivo no dia 200 pós- infecção com Mtb.
[00488]Estes resultados demonstram que a proteção contra a infecção Mtb foi atingida com 3 doses da proteína de fusão ID83 formulada com GLA/CpG-SE. Além disso, a vacinação com ID83 + GLA/CpG-SE protegeu porcos-da-índia Mtb- infectados mais longos do que BCG.
[00489]Em kit, estes resultados indicam que a vacinação com a proteína de fusão ID83 adjuvanted com CpG-SE, GLA-SE, ou CpG/GLA-SE reduziu a carga bac- teriana nos pulmões de camundongos Mtb-infectados, e parcialmente protegeu por- cos-da-índia da infecção por M. tuberculosis. ID83 + CpG/GLA-SE foi a formulação bem mais eficaz em reduzir o número de bactérias viáveis nos pulmões de camundongos Mtb-infectados e prolongar a sobrevivência de porcos-da-índia Mtb- infectados.
[00490]A vacinação de camundongos com três doses de ID83 ou proteína de fusão ID93, adjuvanted com o GLA-SE, induziu o anticorpo e Th1 T respostas de memória de célula junto com a redução de bacilos viáveis inclui o pulmão de camun-dongos infectados com M. tuberculosis. Além disso, observou-se que uma combinação de CpG e GLA-SE era a proteção aumentada bem mais imunogênica e conferida ao desafio de M. tuberculosis.
EXEMPLO 25
RESPOSTAS IMUNES A AD5-ID83 EM CAMUNDONGOS C57BL/6 E PROTEÇÃO CONTRA UM AEROSSOL M DE DESAFIO DE TUBERCULOSE
[00491 ]Este exemplo demonstra que a imunização de camundongos com um vetor de adenovirus projetado para expressar proteínas de fusão ID83 de acordo com a invenção é imunogênica em camundongos C57BL/6.
MATERIAL & MÉTODOS:
Construção de vírus e purificação
Ad5-ID83 foi construído usando o AdEasy® XL
[00492]Sistema de Vetor de Adenoviral (Stratagene #240010). Resumidamente, ID83 foi amplificada do DNA de plasmídio usando PCR, digeriu com HinDIll e EcoRV, e ligar em pShuttle-CMV para fazer ID83-pShuttleCMV. ID83-pShuttleCMV foi linearized digerindo com Pmel e electroporated (2.4kV, 186 Ω, 0.2cm gap cuvette) em Escherichia coli BJ5183-AD-1 células electro-competentes (Stratagene #200157). Os plasmídios Ad5-ID83 recombinantes são identificado digerindo com a Almofada. A almofada digeriu o plasmídio Ad5-ID83 (4pg) foi transfectado em células do ANÚNCIO 239 em placas de 60 mm usando reagente de Polifect (Invitrogen #301107). Depois que as células s de 4 dias são colhido em 3ml_ meio e lisado por três ciclos da geada/degelo. O sobrenadante de lisado foi usado para amplificar o vírus da purificação pela centrifugação de gradiente CsCI. Imunização
[00493]Os camundongos C57/BL6 femininos são obtido de Charles River e idade - combinado (5-7 semana) dentro de cada experimento. Os camundongos são imunizado duas vezes (de 3 semanas à parte) com 5 x 108 partículas virais Ad5- ID83. Os camundongos na salina, e os grupos de controle da BCG receberam PBS ou uma dose única de 5 x 104 BCG CFU respectivamente. Os camundongos são injetado com um volume total de 100pl/mouse via a via i.m..
Citoquina ELISPOT
[00494]A microplaca de filtração de 96 poços tipo MultiScreen (Millipore, Be-dford, Massachusetts) são coberto de 10 pg/mL IFN-y de rato anti-camundongo ou TNF capturam Ab (eBioscience) e incubado durante a noite em 4 °C. Placas foram lavados com PBS, bloqueado com 1640 RPMI e FBS de 10 % de pelo menos 1 h em temperatura ambiente, e lavado novamente. Os esplenócitos são plaqueados em duplicata em 2 x 105 células/poço, e estimulado com o meio, Con A 3 pg/mL, PPD 10 pg/mL, ou cada proteína recombinante 10 pg/mL por 48 h a 37 °C. As placas são posteriormente lavadas com PBS e 0,1 % de Tween-20 e incubado por 2 h com um anti-camundongo de rato conjugado pela biotina IFN-y ou Ab secundário TNF (eBioscience) em 5 pg/mL nPBS, BSA de 0,5 %, e 0,1 % de Tween-20. Os filtros são o utilizando desenvolvido do ALFA Vectastain avidin peroxidase conjugado e Vectastain AEC kits de substrato (Laboratórios de Vetor, Burlingame, Califórnia) de acordo com o protocolo do fabricante. A reação foi parada lavando as placas com a água de deionizada, as placas são secas no escuro, e as manchas são contado so- bre uma automatizou o leitor ELISPOT (C.T.L. Serie3A Analisador, Cellular Technology Ltd, Cleveland, Ohio), e analisado com Immunospot ® (CTL Analisador LLC).
Experimento de proteção
[00495]Os camundongos são s.c imunizado., três vezes, 3 semanas à parte, com 8 pg da proteína de fusão, formulada no adjuvante indicado. Os camundongos de controle seguros são imunizado com BCG (5 x 104 CFU) na base da cauda (uma vez), e os animais de controle negativos são injetado com a salina, ou adjuvante sozinho. S de trinta dias depois da imunização última, os camundongos são provocado pela exposição de aerossol de dose baixa com Mycobacterium tuberculosis cepa de H37Rv (ATCC 35718; Coleção de Cultura de Tipo americana, Manassas, VA) utilizando de uma câmara de exposição de aerossol de UW-MADISON (Madison, Wl) calibrado para liberar 50-100 bactérias nos pulmões. Quatro semanas depois, os camundongos são euthanized, e o pulmão e o baço homogenates foram preparados em PBS/Tween 80 (0.05 %). As contas bacterianas são determinam pela galvanização diluições seriais de órgãos inteiros individuais em Middlebrook nutriente 7H11 Ágar-ágar de Bacto (BD Biosciences, Cockeysville, Maryland) e contagem de formação de colônia bacteriana depois da incubação de 14 dias em 37 °C no ar umedeci- do e CO2de5%. Os dados são expressados como LoglO do número avaro de bactérias recuperou SD ±, e Redução LoglO de CFU = LoglO CFU para o grupo vacinado - LoglO CFU para o grupo tratado Salino.
RESULTADOS:
[00496]As respostas imunes a Ad5-ID83 C57BL/6 camundongos são imunizado duas vezes, três semanas à parte, com Ad5-ID83.
[00497]Três semanas depois da imunização última, os esplenócitos foram preparados e provaram por ELISPOT determinar o número relativo de IFN- - esplenócitos expressam em resposta ao meio sozinho, o mitogen ConA, e cada uma das proteínas de fusão.
[00498]A imunização com Ad5-ID83 induziu o antígeno IFN-específico y res-postas de revocação por esplenócitos ativados (a Figura 7A). A injeção com a salina não induziu IFN-y respostas específicas para ID83.
[00499]A proteção permitida por Ad5-ID83 contra um aerossol desafia com Mtb H37Rv.
[00500]O número de bacilos viáveis, expressados como média LoglO CFU, no pulmão de camundongos vacinados com 5 x 108 partículas virais Ad5-ID83, é determinado 4 semanas põem o desafio de aerossol no correio com-50 CFU de M. tuberculosis virulento H37RV. O média Logw CFU no pulmão de camundongos imu-nizados com Ad5-ID83 foi em comparação com o média LoglO CFU obtido em ca-mundongos que recebem placebo (salina). A diferença no média LoglO CFU no grupo salino contra os grupos vacinados é expressada como redução de LoglO de CFU.
[00501]A imunização de camundongos com duas doses de Ad5-ID83 resultou em uma redução em bacilos Mtb viáveis no pulmão de camundongos Mtb- infectados de 0.27 (a Figura 7B). Estes resultados são promessa naquela proteção contra a infecção Mtb foi atingido com somente 2 doses de Ad5-ID83.
[00502]Em kit, estes resultados indicam que a imunização com Ad5-ID83 in-duziu a célula T respostas imunes e parcialmente protegeu camundongos de um aerossol desafio de M. tuberculosis.
EXEMPLO 26 IMUNOTERAPIA COM MTB RV1813, RV2608, E PROTEÍNAS RECOMBINANTES RV3620 COM O GLA-SE DE ADJUVANTE.
[00503]Este exemplo demonstra que a imunização de camundongos com uma mistura de proteínas recombinantes de acordo com a invenção junto com a terapia antibiótica padrão pode prolongar a vida de M. tuberculosis - infectam camundongos.
MATERIAL & MÉTODOS:
[00504]As proteínas recombinantes e as formulações de adjuvante proteínas Recombinantes são produzido tal como descrito acima.
[00505]O lipídio A de Glicopiranosil (GLA) foi obtido de Avanti (Alabastro, a Alabama). As emulsões de óleo na água estáveis (-SE) foram preparadas.
Desafio de aerossol com tuberculose M.
[00506]Os camundongos SWR/J femininos são obtido de Laboratórios de Jackson e combinado pela idade (5-7 semana) dentro de cada experimento. Os ca-mundongos são provocado pela exposição de aerossol de dose baixa com M. tuber-culosis cepa de H37Rv (ATCC 35718; Coleção de Cultura de Tipo americana, Ma-nassas, VA) utilizando de uma câmara de exposição de aerossol de UW-MADISON (Madison, Wl) calibrado para liberar 50-100 bactérias nos pulmões. A sobrevivência de camundongos foi controlada para a Terapia de pós-infecção s de 225 dias
[00507]Duas semanas depois de um teste de provocação com aerossol com M. tuberculosis, o tratamento antibiótico padrão foi iniciado. Os camundongos são fornecidos 50 mg/l de rifampin e 85 mg/l isoniazide na sua água potável por 60 dias. Os camundongos alguns receberam a imunoterapia adicional e ser imunizado no dia 76, dia 97, e dia 118 pós-infecção com 6 pg de cada Rv1813, Rv2608, e proteína recombinante Rv3620 formulada com 20 pg do GLA-SE de adjuvante. Os camundongos são injetado com um volume total de 100pl/mouse via a via s.c.. A sobrevivência de camundongo foi monitorada por 225 dias.
RESULTADOS: Proteção permitida por uma combinação de antibióticos + Rv1813, Rv2608, e Rv3620 com a imunoterapia de GLA-SE, EM M. tuberculosis - infectam camundongos.
[00508]A sobrevivência de tratado de camundongos Mtb-infectado com um regime padrão de rifampin + isoiazide antibióticos (Rx) ou com uma combinação de Rx + imunização com Rv1813, Rv2608, e proteínas recombinantes Rv3620 formula- das com 20 pg do GLA-SE de adjuvante (imunoterapia) foi seguida para o desafio de aerossol de correio por 225 dias com-50 CFU de M. tuberculosis virulento H37Rv.
[00509]A sobrevivência de tratado de camundongos com Rx + imunoterapia foi em comparação com a sobrevivência de camundongos que recebem Rx sozinho ou placebo (salina).
[00510]O tratamento de camundongos com três doses de Rv1813, Rv2608, e proteínas recombinantes Rv3620 com o GLA-SE, além de Rx, resultou na sobrevivência aumentada de camundongos Mtb-infectados (a Figura 8). Em dia 225 pós- infecção, 75 % do animal vacinado com Rv1813, Rv2608, e Rv3620 com o GLA-SE são ainda vivo, comparado com 0 % dos camundongos no antibiótico (Rx) o tratamento sozinho agrupa-se, e 0 % no grupo de placebo.
[00511]Estes resultados demonstram que a proteção contra a infecção Mtb foi atingida com antibióticos + 3 doses de Rv1813, Rv2608, e Rv3620 com o GLA- SE. Além disso, o tratamento com antibióticos + Rv1813, Rv2608, e Rv3620 com o GLA-SE protegeu camundongos Mtb-infectados mais longos do que antibióticos sozinhos. Em kit, estes resultados indicam que imunoterapia com Rv1813, Rv2608, e Rv3620 com o GLA-SE junto com antibióticos induziram respostas imunes que ajudaram camundongos a controle uma infecção de M. tuberculosis estabelecida.
EXEMPLO 27 DIAGNÓSTICO SOROLÓGICO DE TUBERCULOSE
[00512]Este exemplo identifica antígenos de M. tuberculosis e fusões de an-tígeno que tendo aumentam sensibilidade e especificidade do diagnóstico sorológico da infecção por tuberculose.
[00513]As microplacas de 96 poços de Polisorp (Nunc, Rochester, Nova Iorque) são coberto de 2 pg/mL antígeno recombinante no tampão de bicarbonato durante a noite em 4 °C e bloqueado durante 2 horas na temperatura ambiente com PBST com 1 % (p/v) BSA sobre uma batedeira de placa. O soro é diluído apropriadamente a 1/200 em PBST com BSA de 0.1 %, acrescentado a cada um poço e as placas são incubado na temperatura ambiente durante 2 horas com a sacudidela. As placas foram lavados com PBST com o e depois de BSA de 0.1 % HRP conjugou a imunoglobulina IgG (Sigma, St. Louis, o Missouri), diluído 1:10000 em PBST e BSA de 0.1 %, foram acrescentadas a cada um poço e incubaram na temperatura ambiente durante 60 minutos com a sacudidela. Depois da lavagem, as placas são desenvolvido com o substrato de cor de peroxidase (KPL, o Baltimore MD) com a reação extinta pela adição de 1 N H2SO4 depois de 10 minutos. A densidade ótica corrigida de cada um poço em 450-570nm foi lida usando um VERSAmax ® leitor de microplaca (Dispositivos Moleculares, Sunnyvale, Califórnia).
[00514]Os resultados destes experimentos são resumidos em Figuras 9. Uma lista da saliva positiva, a tuberculose confirmou amostras de soro (TB, N = 80 a 92) e uma lista da tuberculose que soro de controle negativo, são (NEC, N = 40 a 46) são analisado para a reatividade com antígenos de tuberculose selecionados. Um antígeno anteriormente caracterizado, TBF10, foi usado como um controle positivo e encontrado para fornecer respostas soropositivas a 53 das 92 amostras soro positivas de tuberculose. A reatividade de antígenos individuais é mostrada em Figuras 9, com todos os antígenos listados expondo reatividade a 11-82 das amostras de soro de tuberculose, com baixo ou nenhuma reatividade ao controle são. A reatividade de um antígeno fornecido variado ao soro apainela de tal modo que as respostas positivas de 100 % possam ser obtidas pela seleção de combinações de antígeno próprias. EXEMPLO 28 CLONAGEM E EXPRESSÃO DA RV0577 RECOMBINANTE Usando DNA genômico H37Rv como padrão, Rv0577 foi amplificada por PCR utilizando os seguintes iniciadores: 5’-RvO577-Ndel CAATTACA LAFGAGAGTTTTGTTGCTGGGACCG (SEQ ID NO: 295) 3'RvO577-Hindlll- CAATTAAAGCTrCTACTTTCCAGAGCCCGCAACGC (SEQ ID NO: 296)
[00515]A amplificação foi realizada sob as seguintes condições: 94 °C 0,5 minutos, 55 °C 0,5 rnin., 68 °C 2 min de 30 ciclos, para fornecer o produto apresentado na SEQ ID N °: 185. O produto do PCR foi digerido com Ndel / Hindlll e clonado no vetor pET28.a. Rv0733 foi expressado por BL-21 de cepa de hospedeiro plysS. O sobrenadante esteve ligado com a resina Ni sob condições de desnaturação. A puri-ficação Ni-NTA foi seguida por uma purificação de troca aniônico. Dializadas em 20 pH de Tris de mm 8.. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 186. EXEMPLO 29 CLONAGEM E EXPRESSÃO DA RV1626 RECOMBINANTE Usando DNA genômico H37Rv como padrão, Rv1626 foi amplificada por PCR utilizando os seguintes iniciadores: 5’-Rv1626-Ndel CAATTAC PAT GACCGGCCCCACCACCGCGCC (SEQ ID NO: 297) 3’-Rv1626-Hindlll CAATTAAAGC77TCAGGTGTCTTTGGGTGTTCCGAG (SEQ ID NO. 298)
[00516]A amplificação foi realizada sob as seguintes condições: 94 °C 0,5 minutos, 55 °C 0,5 minutos, 68 °C 2 min de 30 ciclos, para fornecer o produto apre-sentado na SEQ ID N °: 188. O produto do PCR foi digerido com Ndel / Hindlll e clonado no vetor pET28.a. Rv1626 foi expressado por BL-21 de cepa de hospedeiro plysS. O sobrenadante esteve ligado com a resina Ni sob condições de desnaturação. A purificação Ni-NTA foi seguida por uma purificação de troca aniônico. Dializadas em 20 pH de Tris de mm 8. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 189.
EXEMPLO 30
CLONAGEM E EXPRESSÃO DA RV0733 RECOMBINANTE
[00517]Usando DNA genômico H37Rv como padrão, Rv0733 foi amplificada por PCR utilizando os seguintes iniciadores: 5’-RvO733-5Ndel CAATTA C A TA 7GAGAGTTTTGTTGCTGGGACCG (SEQ ID NO: 299) 3’-RvO733-Hindlll CAATTAA4GCT7CTACTTTCCAGAGCCCGCAACGC (SEQ ID NO: 300) Amplificação foi realizada sob as seguintes condições: 94 °C 0,5 minutos, 55 °C 0,5 minutos, 68 °C 2 min de 30 ciclos, para fornecer o produto apresentado na SEQ ID NO: 191. O produto do PCR foi digerido com Ndel / Hindlll e clonado no vetor pET28.a. Rv0733 foi expressado por BL-21 de cepa de hospedeiro plysS. O sobre- nadante esteve ligado com a resina Ni sob condições de desnaturação. O Ni-NTA purificação foi seguido por uma purificação de troca aniônico. Dializadas em 20 pH de Tris de mm 8. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 192.
EXEMPLO 31 CLONAGEM E EXPRESSÃO DA RV2520 RECOMBINANTE
[00518]Usando DNA genômico H37Rv como padrão, Rv2520 foi amplificada por PCR utilizando os seguintes iniciadores: 5’-Rv2520-Ndel-6his CAATTACA TA 7GCATCACCATCACCATCACGTGGTGGACCGC GATCCCAATACC (SEQ ID NO: 301) 3’-Rv2520-EcoRI CAATTAGA47TCTCAG CG ATTCCTG ATCTTGTG (SEQ ID NO: 302) A amplificação foi realizada sob as seguintes condições: 94 °C 0,5 minutos, 55 °C 0,5 minutos, 68 °C 2 min de 30 ciclos, para fornecer o produto apresentado na SEQ ID N °: 194. O produto do PCR foi digerido com Ndel / EcoRI e clonado em um animal modificado 28a ausência o à montante 6 histidina e o 5'linker sequência. Rv2520 foi transformado em BL-21pl_ysS de hospedeiros de expressão e Rosetta pLysS. Ambas expressado igualmente, mas prosseguido o BL-21 pLysS cepa de célula. Seguinte lise de célula, a fração de sobrenadante esteve atada com a resina Ni-NTA sob condições de desnaturação. A purificação Ni-NTA foi seguida por uma purificação de troca aniônico. As frações purificadas são dializadas em 20 pH de Tris de mm 8. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 195.
EXEMPLO 32
CLONANDO-SE E EXPRESSÃO DA RV1253 RECOMBINANTE
[00519]Usando DNA genômico H37Rv como padrão, Rv1253 foi utilizando amplificado por PCR dos seguintes iniciadores: 5’-Rv1253-Ndel CTGGATCCC EAT GGCCTTCCCGGAATATTCGC (SEQ ID N °: 303) 3’-Rv1253-EcoRI CTAGCTGWTCTCATCCGACGTGTTTCCGCCG (SEQ ID NO: 304)
[00520]A amplificação foi realizada sob as seguintes condições: 94 °C 0,5 minutos, 55 °C 0,5 minutos, 68 °C 2 min de 30 ciclos, para fornecer o produto apre-sentado na SEQ ID N °: 197. O produto do PCR foi digerido com Ndel / EcoRII e clonado no vetor pET28.a. Rv1511 foi transformado no hospedeiro de expressão Rosetta plysS. Depois do lise de um 1L indução, a proteína recombinante foi expressada na pelota de corpo de inclusão. A purificação de afinidade de Ni-NTA foi feita sob condições de desnaturação, depois dializada contra 20 pH de Tris de mm 8.0. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 198.
EXEMPLO 33
CLONAGEM E EXPRESSÃO DA RV1980 RECOMBINANTE
[00521]Usando DNA genômico H37Rv como padrão, Rv1980 foi amplificada por PCR utilizando os seguintes iniciadores: 5’-Rv1980-Ndel-24 CAATTA CA TA 7GGCGCCCAAGACCTACTGCGAG (SEQ ID N °: 305) 3’-Rv1980-Hindlll O CMTTA A GC77OTAGGCCAGCATCGAGTCGATCGC (SEQ ID NO: 306) Amplificação foi realizada sob as seguintes condições: 94 °C 0,5 minutos, 5 δ °C 0,5 minutos, 68 °C 2 min de 30 ciclos, para fornecer o produto apresentado na SEQ ID NO: 200. O produto do PCR foi digerido com Ndel / Hindlll e clonado no vetor pET28.a. Rv1980 foi transformado no hospedeiro de expressão Rosetta plysS. Depois do lise de um 1L indução, a proteína recombinante foi expressada na pelota de corpo de inclusão. A purificação de afinidade de Ni-NTA foi feita sob condições de desnaturação, depois dializada contra 20 pH de Tris de mm 8.0. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 201. EXEMPLO 34 CLONAGEM E EXPRESSÃO DA RV3628 RECOMBINANTE Usando DNA genômico H37Rv como padrão, Rv3628 foi amplificada por PCR utilizando os seguintes iniciadores: 5’-Rv3628-Nde-6hisl CAATTACA TA 7GCATCACCATCACCATCACATGCAATTCGACG TGACCATC (SEQ ID NO: 307) 3’-Rv3628-EcoRI CAATTAGA47TCTCAGTGTGTACCGGCCTTGAAGCG (SEQ ID NO: 308)
[00522]A amplificação foi realizada sob as seguintes condições: 94 °C 0,5 minutos, 55 °C 0,5 minutos, 68 °C 2 min de 30 ciclos, para fornecer o produto apresentado na SEQ ID NO: 203. Usando DNA genômico H37Rv como padrão, Rv3628 foi PCR'd com condições 95 °C 1 minuto, 58 °C 1 minuto, 72 °C 1.5 min de 35 ciclos. O produto do PCR foi digerido com Ndel / EcoRI e clonado na pET 17b. Rv3628 foi transformada nos hospedeiros de expressão BL-21 plysE e Rosetta plysS. Ambas expressado igualmente, mas prosseguido o construtor de plysE. Depois da lise de 1 L da solução de indução, ele entrou no corpo de inclusão. Ni-NTA foi feito sob condições de desnaturação, depois dializada contra 10 mM de Tris em pH 8,0. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 204. EXEMPLO 35 CLONANDO-SE E EXPRESSÃO DA RV1844 RECOMBINANTE Usando DNA genômico H37Rv como padrão, Rv1844 foi utilizando amplificado por PCR dos seguintes iniciadores: 5’-Rv1884-Ndel-6his30 CAATTACATATGCATCACCATCACCATCACACTTCCGGCGAT ATGTCGAGC (SEQ ID NO: 309) 3’-Rv1884-EcoRI CAATTAGAA rrCTCAGCGCGGAATACTTGCCTG (SEQ ID N °: 310)
[00523]A amplificação foi realizada sob as seguintes condições: 94 °C 0.5min., 55 °C 0,5 minutos, 68 °C 2 min de 30 ciclos, para fornecer o produto apresentado na SEQ ID NO: 206. O produto do PCR foi digerido com Ndel / EcoRI e clonado na pET 17b. O plasmídio contendo o gene Rv1884 foi transformada nos hospedeiros de expressão BL-21 plysE e plysS. Ambas expressado igualmente, mas prosseguido o plysE. Depois da lise de 1 L da solução de indução, permaneceu na fração de corpo de inclusão insolúvel. Ni-NTA foi feito sob condições de desnaturação, depois dializada contra 10 mM de Tris em pH 8,0. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 207.
EXEMPLO 36 CLONAGEM E EXPRESSÃO DA RV3872 RECOMBINANTE
[00524]Usando DNA genômico H37Rv como padrão, Rv3872 foi amplificada por PCR utilizando os seguintes iniciadores: 5’-Rv3872-Ndel GTGCTAGCC A TA 7GGAAAAAATGTCACATGATC (SEQ ID NO: 311) 3’-Rv3872-Hindlll CTGGATCC A GC77CTATTCGGCGAAGACGCCGGC (SEQ ID NO: 312) A amplificação foi realizada sob as seguintes condições: 94 °C 0,5 minutos, 55 °C 0,5 minutos, 68 °C 2 min de 30 ciclos, para fornecer o produto apresentado na SEQ ID NO: 209. O produto do PCR foi digerido com Ndel / Hindlll e clonado no vetor pET28.a. Rv3872 foi transformado no hospedeiro de expressão Rosetta plysS. Depois da lise de 1 L da solução de indução, a proteína recombinante foi expressada na fração de sobrenadante solúvel. A purificação de afinidade de Ni-NTA foi feita 2X condições sob nativas, depois dializada contra 20 pH de Tris de mm 8.0. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 210. EXEMPLO 37 CLONANDO-SE E EXPRESSÃO DA RV3873 RECOMBINANTE Usando DNA genômico H37Rv como padrão, Rv3873 foi utilizando amplificado por PCR dos seguintes iniciadores: 5’-Rv3873-Ndel GTGCTAGCC/AT/ArGCTGTGGCACGCAATGCCAC (SEQ ID NO: 313) 3’- 3873-Hindlll CTGGATCCA4GC IT CACCAGTCGTCCTCTTCGTC (SEQ ID N°: 314)
[00525]A amplificação foi realizada sob as seguintes condições: 94 °C 0,5 minutos, 55 °C 0,5 minutos, 68 °C 2 min de 30 ciclos, para fornecer o produto apre-sentado na SEQ ID NO: 212. O produto do PCR foi digerido com Ndel / Hindlll e clonado no vetor pET28a. O plasmídio contendo o gene Rv3873 foi transformado no hospedeiro de expressão Rosetta plysS. Depois da lise de 1 L da solução de indução, a proteína recombinante foi expressada na fração de sobrenadante solúvel. O Ni-NTA purificação de afinidade foi feito 2X condições sob nativas, depois dializada contra 20 pH de Tris de mm 8.0. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 213. EXEMPLO 38 CLONAGEM E EXPRESSÃO DA RV1511 RECOMBINANTE Usando DNA genômico H37Rv como padrão, Rv1511 foi amplificada por PCR utilizando os seguintes iniciadores: 5’-Rv1511-Ndel CAATTA CA TA 7GCATCACCATCACCATCACGTGAAGCGAGCG CTCATCACC (SEQ ID NO: 315) 3’-Rv1511-EcoRI CAATTAGAA 7TCTCATGTCCGGCCGGCGATCATCG (SEQ ID N°: 316)
[00526]A amplificação foi realizada sob as seguintes condições: 94 °C 0,5 minutos, 55 °C 0,5 minutos, 68 °C 2 min de 30 ciclos, para fornecer o produto apre-sentado na SEQ ID NO: 214. O produto do PCR foi digerido com Ndel / EcoRI e clonado na pET 28a, menos o 5' ligante. Rv1511 foi transformado em BL-21 plysS de hospedeiros de expressão e Rosetta plysS. Ambas expressado igualmente, mas prosseguido as células BL-21. Depois da lise de 1 L da solução de indução, a proteína recombinante foi expressada na pelota de corpo de inclusão. A purificação de afinidade de Ni-NTA foi feita sob condições de desnaturação, depois dializada contra 10 pH de Tris de mm 9.5. A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 215. EXEMPLO 39 CLONAGEM E EXPRESSÃO DA PROTEÍNA DE FUSÃO RECOMBINANTE ID93 Os seguintes iniciadores são usadas em para clonagem a fusão constroem ID93, que compreende os parceiros de fusão derivaram de Rv3619, Rv1813, Rv3620 e Rv2608: 5': Rv1813mat-5Ndel-Kpnl CAATTACyA TA 7GGG7> ACCCATCTCGCCAACGGTTCGATG (SEQ ID NO: 218) 3': Rv1813mat-3Saclgo CAATTAGAGCTCGTTGCACGCCCAGTTGACGAT (SEQ ID N °: 219) 5': Rv3620-5Sacl CAATTAG A GCrCATGACCTCGCGTTTTATGACG (SEQ ID NO: 220) 3': Rv3620-3Sallgo CAATTAG7CG A CGCTGCTGAGGATCTGCTGGGA (SEQ ID NO: 221) 5': Rv2608-5Sall CAATTAGrCGACATGAATTTCGCCGTTTTGCCG (SEQ ID NO: 222) 3': Rv2608-3Scal-Hindlll CAATTAAAGC77TTMGLAC7OAAAAGTCGGGGTAGCGCCGG (SEQ ID NO: 223) 51: Rv3619-5Ndel CAATTACyA TA 7GACCATCAACTATCAATTC (SEQ ID NO: 224) 3': Rv3619-3Kpnl CAATTAGGTACCGGCCCAGCTGGAGCCGACGGC (SEQ ID NO: 225)
[00527]Rv1813 e Rv3620 são amplificadas por PCR padrão do DNA genômico H37Rv (94 °C para 0:30; 58 °C para 0:30; 58 °C para 1:30; 35 ciclos). Rv1813 foi digerido com Ndel/Sacl então clonado no vetor pET28.a. Rv3620 foi digerido com Sacl/Sall então ligam no construtor de Rv1813pET. O construtor de fusão mandou apresentar uma sequência de polinucleotídeo em SEQ ID NO: 217, codificando a proteína de fusão apresentada na SEQ ID NO: 226. Rv2608 foi amplificada do padrão de plasmídio por PCR (94 °C para 0:30; 58 °C para 0:30; 68 °C para 1:30; 35 ciclos). O produto foi digerido com a Vela / Hindlll e clonado em pet28.a-
[00528]Vetor de Rv1813-3620. Rv3619 foi amplificada mesmo como acima e digerido com Ndel / Kpnl então ligam no vetor ID83. ID93 foi expressado no hospedeiro BL-21 plysS (2L, 2XYT meio de crescimento, 37 °C). Induzido com 1 mM IPTG em OD 0.77 e colhido em OD 1.93. A pelota de célula foi suspensa no tampão de lise (20 Tris de mm pH8, 100 mM NaCI, 2 mM PMSF) e congelou-se. A pelota de célula então foi descongelada, lisada por ultrassom, e girada em 7,000rcf durante 20 minutos ID83 é uma proteína de corpo de inclusão. A pelota foi lavada 2x com Sujeitos de 1 %. A pelota foi solubilized em 60 ml_ no tampão obrigatório (8M uréia, 20 pH de Tris de mm 8, 100 mM NaCI) e ligou a 16 ml_ Ni-NTA a resina na TEMPERATURA AMBIENTE durante 1 hora. A resina foi lavada (50 DOUTOR de 0,5 % ml_ durante 20 minutos; 80 IPA de 60 % ml_ durante 30 minutos, 50 ml_ 0.5%DOC enxaguadura) o e depois eluído com o tampão obrigatório com imidazol a 300 mM. O sobrenadante desde o princípio liga esteve ligado a uma 8 resina ml_ adicional e processado como indicado acima. As purificações acima mencionadas removeram produtos de emergência. Outros Ni-NTA ligam foi feito durante a noite em 4 °C em 160 ml_ (tampão obrigatório com 50 mM NaCI) com 32 resina ml_. A resina foi lavada e eluído como indicado acima. As frações disto ligam são dializadas em 20 Tris de mm pH8.
EXEMPLO 40 CLONAGEM E EXPRESSÃO DA PROTEÍNA DE FUSÃO RECOMBINANTE ID91
[00529]Os seguintes iniciadores são usadas em para clonagem a fusão constroem ID91, que compreende os parceiros de fusão derivaram de Rv3619, Rv2389, Rv3478 e Rv1886: 5’-Rv3619-5Ndel CAATTAC A TA 7GACCATCAACTATCAATTC (SEQ ID NO: 228) 3’-Rv3619-3Kpnl CAATTAGGLACCGGCCCAGCTGGAGCCGACGG (SEQ ID NO: 229) 5’-Rv2389-Kpnl TGGGCCGGLACCGACGACATCGATTGGGACGCC (SEQ ID N°: 230) 3’-Rv2389-BamHI AATCCACCAC GGA CCATCGTCCCTGCTCCCCGAAC (SEQ ID NO: 231) 5’-Rv3478-BamHI CAGGGACGATGG T CCGTGGTGGATTTCGGGGCGTTAC (SEQ ID NO: 232) 3’-Rv3478-EcoRI CCGGGAGAA GAA 77CTCCGGCGGCCGGTGTGCGGG (SEQ ID NO: 233) 5’-Rv1886-EcoRI GCCGCCGGAG A 77CTTCTCCCGGCCGGGGCTGCC (SEQ ID N°: 234) 3’-Rv1886matR Hindlll GATATO V\GC7TTCAGCCGGCGCCTAACGAAC (SEQ ID N °:235)
[00530]Q construtor de fusão mandou apresentar uma sequência de polinu-cleotídeo em SEQ ID NO: 227, codificando a proteína de fusão apresentada na SEQ ID NO: 236.
EXEMPLO 41 CLONAGEM E EXPRESSÃO DA PROTEÍNA DE FUSÃO RECOMBINANTE ID71
[00531]Os seguintes iniciadores são usadas em para clonagem a fusão constroem ID71, que compreende os parceiros de fusão derivaram de Rv3619, Rv2389, Rv3478 (N180) e Rv1886: 5’-Rv3619-5Ndel CAATTA CA TA 7GACCATCAACTATCAATTC (SEQ ID NO: 238) 3’-Rv3619-3Kpnl CAATTAGG77\CCGGCCCAGCTGGAGCCGACGG (SEQ ID N °: 239) 5’-Rv2389-Kpnl TGGGCCGGTACCGACGACATCGATTGGGACGCC (SEQ ID N°: 240) 3’-Rv2389-BamHI AATCCACCACGG A rCCATCGTCCCTGCTCCCCGAAC (SEQ ID N°: 241) 5’-Rv3478-N180-EcoRI (SEQ ID NO: 242) 5’-Rv1886-EcoRI GCCGCCGGAGA4 T CTTCTCCCGGCCGGGGCTGCC (SEQ ID NO: 243) 3’-Rv1886matR Hindlll GATATC A GC77TCAGCCGGCGCCTAACGAAC (SEQ ID NO: 244)
[00532]0 construtor de fusão mandou apresentar uma sequência de polinucleotídeo em SEQ ID NO: 237, codificando a proteína de fusão apresentada na SEQ ID NO: 245.
EXEMPLO 42 CLONAGEM E EXPRESSÃO DA PROTEÍNA DE FUSÃO RECOMBINANTE ID114
[00533]Os seguintes iniciadores são usadas em para clonagem a fusão constroem ID114, que compreende os parceiros de fusão derivaram de Rv1813, Rv3620, Rv2608 e Rv1886: 5': Rv2608-5Sall CAATTAG7CGACATGAATTTCGCCGTTTTGCCG (SEQ ID NO: 247) 3':Rv2608-3Scal- HindlllCAATTAAAGCmTAAGTACTGAAAAGTCGGGGTAGCGCCGG (SEQ ID NO: 248) 5’-Rv1886-2608-Scal CGGCGCTACCCCGACTTTTC Gr CTTTCTCCCGGCCGGGG CTGCCG (SEQ ID NO: 249) 3’-Rv1886matR Hindlll GATATC A GCπTCAGCCGGCGCCTAACGAAC (SEQ ID NO: 250) Rv1813 e Rv3620 são amplificadas por PCR do DNA genômico H37Rv de padrão (94 °C para 0:30; 58 °C para 0:30; 58 °C para 1:30; 35 ciclos). O construtor de fusão mandou apresentar uma sequência de polinucleotídeo em SEQ ID NO: 246, codificando a proteína de fusão apresentada na SEQ ID NO: 251. EXEMPLO 43 CLONAGEM E A EXPRESSÃO DA PROTEÍNA DE FUSÃO RECOMBINANTE ID125 os seguintes iniciadores são usadas em para clonar o construtor de fusão ID125, que compreende parceiros de fusão derivou de Rv3619, Rv1813, Rv3620, Rv2608 e Rv1886: 5': Rv2608-5Sall CAATTAG7CGΔCATGAATTTCGCCGTTTTGCCG (SEQ ID NO: 253) 3': Rv2608-3Scal-Hindlll CAATTA/MGC 7T A G C GAAAAGTCGGGGTAGCGCCGG (SEQ ID NO: 254) 5’-Rv1886-2608-Scal CGGCGCTACCCCGACTTTTC G7 C7TTCTCCCGGCCGGGG CTGCCG (SEQ ID NO: 255) S'-Rvi δ ematR Hindlll GATATC A GC IT CAGCCGGCGCCTAACGAAC (SEQ ID NO: 256) O construtor de fusão mandou apresentar uma sequência de polinucleotídeo em SEQ ID NO: 252, codificando a proteína de fusão apresentada na SEQ ID NO: 257. EXEMPLO 44 CLONAGEM E A EXPRESSÃO DA PROTEÍNA DE FUSÃO RECOMBINANTE DID85 os seguintes iniciadores são usadas em para clonar o construtor de fusão DID85, que compreende parceiros de fusão derivou de Rv2032, Rv2875, e Rv0831: 5’-Rv2032-Ndel-6his GATACAC A TA rGCACCATCACCATCACCACATGCCGGACACC ATGGTGAC (SEQ ID NO: 259) 3’-Rv2032-GGSGGS-BamHI GATO GGA CCGCTACCGCCAGAACCACCCCGGTGATCCTTAG CCCGAAC (SEQ ID NO: 260) 5’-Rv2875-BamHI GGTGGTTCTGGCGGTAGCGG/ArrCATGGGCGATCTGGTGAG CCCG (SEQ ID NO: 261) 3’-Rv2875R-EcoRI CATGAA 7TCAGAACCGCCGCTTCCGCCCGCCGGAGGCATTA GCACGC (SEQ ID NO: 262) 5’-RvO831 F-EcoRI GGCGGAAGCGGCGGTTCTGA4 TCATGCTCCCCGAGACAAA ICAG (SEQ ID NO: 263) 3’-RvO831 R-Hiπdlll TAGAATTCMGC7TTTACTGGCGAAGCAGCTCATC (SEQ ID NO: 264)
[00534]Os genes de Rv2032, Rv2875, e Rv0831 são amplificadas por PCR do DNA de Plasmidio existente (94 °C para 0:30; 58 °C para 0:30; 58 °C para 1:30; 30 ciclos) utilizando das sequências acima mais principais. Os produtos PCR amplificados dos três são usadas em um segundo círculo de PCR para amplificar o produto genético de fusão de comprimento cheio usando os 5’-Rv2032-Ndel-6his e 3’- Rv0831 R-Hindlll iniciadores. O produto do PCR resultante foi digerido com Ndel / hindi 11 e clonado no vetor pET29a. DID85 foi expressado por BL-21 de cepa de hospedeiro plysS. O construtor de fusão mandou apresentar uma sequência de polinucleotídeo em SEQ ID NO: 258, codificando a proteína de fusão apresentada na SEQ ID NO: 265. Depois da lise de 1 L da solução de indução, ele entrou no corpo de inclusão. Ni-NTA foi feito sob condições de desnaturação, seguidas pela croma- tografia de troca aniônico. As frações purificadas são dializadas contra 10 mM de Tris em pH 8,0. EXEMPLO 45 CLONAGEM E EXPRESSÃO DA PROTEÍNA DE FUSÃO RECOMBINANTE DID92 Os seguintes iniciadores são usadas em para clonagem a fusão constroem DID92, que compreende os parceiros de fusão derivaram de Rv3044, Rv1009, e Rv0614: 5’-Rv3044-Ndel-6his G ATACAC A TA 7GCACCATCACCATCACCACATGGGCA GCAGC CATCATCATC (SEQ ID NO: 267) 3’-Rv3044-Ncol CATATCGAGC7CGTTGATCGGCGCGTCGACCC (SEQ ID NO: 268) 5’- Rv1009-Ncol-GGSGGS ligante ATCAACG/AGCrCGGAGGTTCTGGTGGAAGCGCATGCAAAAC GGTGACGTTGAC (SEQ ID NO: 269) 3’-Rv1009-EcoRI CATATCG AIT CGCGCGCACCCGCTCGTGCAGC (SEQ ID NO: 270) 5 ligante '-Rv0164-EcoRI-GGSGGS CATGTCGAA irCGGTGGAAGCGGAGGTTCTATGACGGCAAT CTCGTGCTCAC (SEQ ID NO: 271) 3’-RvO164-Hindlll CATATC A GC77TTAGCTGGCCGCCAGCTGCTC (SEQ ID NO: 272)
[00535]Q construtor de fusão mandou apresentar uma sequência de polinu-cleotídeo em SEQ ID NO: 266, codificando a proteína de fusão apresentada na SEQ ID NO: 273. EXEMPLO 46 CLONAGEM E EXPRESSÃO DA PROTEÍNA DE FUSÃO RECOMBINANTE DID108 Os seguintes iniciadores são usadas em para clonagem a fusão constroem DID108, que compreende os parceiros de fusão derivaram de Rv3872, Rv3873, Rv3875 e Rv3881: 5’-Rv3872-Ndel-6his GATACAC/*TA7GCACCATCACCATCACCACATGGAAAAAATG TCACATGATC (SEQ ID NO: 275) 3’-Rv3872-Sacl GATACAT G/AGCrCTTCGGCGAAGACGCCGGCGGC (SEQ ID NO: 276) 5 ligante '-Rv3873-Sacl-GGSGGS GATACAG/AGC7CGGAGGTTCCGGTGGAAGCATGCTGTGGCA CGCAATGCC (SEQ ID NO: 277) 3’-Rv3873-EcoRI GATACAG A 7TCCCAGTCGTCCTCTTCGTCCCAG (SEQ ID NO: 278) 5 ligante ‘-Rv3875-EcoRI-GGSGGS GACAGyAyA 7TCGGTGGCAGTGGAGGATCTATGACAGAGCAGC AGTGGAAT (SEQ ID NO: 279) 3’-Rv3875-Nhel CATATCAGCTyAGC TGCGAACATCCCAGT GACGTTG (SEQ ID NO: 280) 5 ligante '-Rv3881-Nhel-GGSGGS CATATCAGCLAGCGGAGGTTCCGGTGGAAGCATGACGCAGT CGCAGACCGTG (SEQ ID NO: 281) 3’-Rv3881 -Hindlll CATATCA A GCTFTCACTTCGACTCCTTACTGTC (SEQ ID NO: 282)
[00536]0 construtor de fusão mandou apresentar uma sequência de polinucleotídeo em SEQ ID NO: 274, codificando a proteína de fusão apresentada na SEQ ID NO: 283. EXEMPLO 47. CLONAGEM E EXPRESSÃO DA PROTEÍNA DE FUSÃO RECOMBINANTE DID93 Os seguintes iniciadores são usadas em para clonagem a fusão constroem DID93, que compreende os parceiros de fusão derivaram de Rv1099, Rv0655, e Rv0054: 5’-Rv1099-Ndel TAGGATCCCyA TA 7GGAGCTGGTCCGGGTGACC (SEQ ID NO: 285) 3 ligante '-Rv1099-EcoRI-GGSGGS CACGM 7TCGCTTCCACCAGAACCTCCGGGCAATGGGTACA CGGCGC (SEQ ID NO: 286) 5 Ligante '-Rv0655-EcoRI-GGSGGS GGAGGTTCTGGTGGAAGCGyA rrCGTGCGATACAGTGACTC ATAC (SEQ ID NO: 287) 3’-RvO655-Sacl GCCACG GC7CAGAACCGCCGCTTCCACCCTGGCCGATTTC GTGCACCGC (SEQ ID NO: 288) 5 ligante '-Rv0054-Sacl-GGSGGS GCCAGGGTGGAAGCGGCGGTTCTGAGCrCGTGGCTGGTGA CACCACCATC (SEQ ID NO: 289) 3'Rv0054-Hindlll CAATTAA4GC77TCAGAATGGCGGTTCGTCATCGCC (SEQ ID NO: 290) O construtor de fusão mandou apresentar uma sequência de polinucleotídeo em SEQ ID N °:284, codificando a proteína de fusão apresentada na SEQ ID NO: 291 EXEMPLO 48 CLONANDO-SE E EXPRESSÃO DA PROTEÍNA DE FUSÃO RECOMBINANTE RV3875 Usando DNA genômico H37Rv como padrão, Rv3875 foi utilizando amplificado por PCR dos seguintes iniciadores: 5’-Rv3875-6His-Ndel CCATTACyA TA 7GCATCACCATCACCATCACATGACAGAGCAG CAGTGGAA (SEQ ID NO: 317) 3’-Rv3875-EcoRI CCATTAG Arr CCTATGCGAACATCCCAGTGAC (SEQ ID NO: 318)
[00537]A sequência de aminoácidos da proteína recombinante é apresentada na SEQ ID NO: 294.
[00538]Estas e outras modificações podem ser feitas a modalidades na luz da descrição acima detalhada. Em geral, nas seguintes reivindicações, os termos usados não devem ser interpretados para limitar as reivindicações para modalidades específicas divulgadas no Relatório Descritivo e as reivindicações, mas devem ser interpretados para incluir todas as modalidades possíveis junto com o alcance cheio de equivalentes aos quais tais reivindicações têm direito. Consequentemente, as reivindicações são não limitadas pela divulgação.