-
Die
Erfindung betrifft eine Nukleinsäure
codierend für
ein nicht fluoreszierendes, aber proteolytisch zur Fluoreszenz aktivierbares
Protein, welches an seinem N-Terminus mindestens eine ein- oder
beidseitig von Protease-Schnittstellen flankierte Stör-Aminosäuresequenz
umfasst, sowie das von dieser Nukleinsäure kodierte Protein oder eines
seiner Fusionsproteine.
-
Die
Erfindung betrifft weiterhin eine Nukleinsäure codierend für ein nicht
fluoreszierendes, aber proteolytisch zur Fluoreszenz aktivierbares
Protein, welches mindestens eine ein- oder beidseitig von Protease-Schnittstellen
flankierte Stör-Aminosäuresequenz
umfasst, die im Protein nach der Aminosäure mit der Position 1, 2,
3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, gezählt vom N-Terminus des Proteins,
insertiert ist, sowie das von dieser Nukleinsäure kodierte Protein oder eines
seiner Fusionsproteine.
-
In
einem weiteren Aspekt, betrifft die Erfindung einen Vektor, der
eine der genannten Nukleinsäuren umfasst,
sowie einen Kit, der einen dieser Vektoren bzw. ein Protein, welches
von einem dieser Vektoren kodiert wird, umfasst.
-
Weiterhin
umfasst die Erfindung ein Verfahren zur Detektion oder zur Charakterisierung
einer Protease-Aktivität
in einer Zelle umfassend die folgenden Verfahrensschritte:
- a) Transfektion einer Zelle
mit einem
rekombinanten Vektor, kodierend für ein nicht fluoreszierendes,
aber proteolytisch zur Fluoreszenz aktivierbares Protein, welches
mindestens eine ein oder beidseitig von Protease-Schnittstellen
flankierte Stör-Aminosäuresequenz
am N-Terminus angehängt
oder durch Insertion umfasst,
- b) Aktivierung einer bislang inaktiven Protease in der Zelle
oder Aktivierung der Expression einer Protease in der Zelle, wobei
die Protease-Schnittstellen aus a) Substrate dieser Protease darstellen,
- c) Generierung eines fluoreszierenden Proteins durch proteolytische
Entfernung der mindestens einen ein- oder beidseitig von Protease-Schnittstellen
flankierten Stör-Aminosäuresequenz
aus a) aus dem nicht fluoreszierenden Protein aus a),
- d) Detektion der Fluoreszenz des fluoreszierenden Proteins aus
c).
-
Der
Abbau bzw. die Spaltung von Proteinen in Organismen wird durch die
Enzymfamile der Proteasen katalysiert. In den letzten Jahren zeigte
sich, dass Proteasen über
den reinen Proteinabbau hinaus ein weitreichendes Aufgabenspektrum
in zellulären
Systemen übernehmen.
Zahlreiche Proteasen haben intrazellulär lokalisierte Substrate. So
spielt beispielsweise das Proteasom, ein Multienzymkomplex, eine
zentrale Rolle beim Abbau von Ubiquitin-markierten Proteinen. Auch
apoptotische Signalkaskaden werden von Proteasen, den Caspasen und
Calpainen initiiert und kontrolliert. Weiterhin sind proteolytische
Aktivitäten
zur Regulation des Zellzyklus notwendig, die Aktivität der Separase.
Extrazelluläre
bzw. sezernierte Proteasen regulieren beispielsweise Prozesse in
der Embryonalentwicklung (Romboid), sind für Auswachsen von Zellfortsätzen von Bedeutung
(insbesondere Metalloproteasen), dienen der Aktivierung von Enzymen
und Signalkaskaden (insbesondere Plasminogen, Thrombin und Renin)
oder katalysieren die Nahrungsverdauung (insbesondere Trypsin, Chymotrypsin).
-
Darüber hinaus
sind Proteasen auch wichtige Angriffspunkte für pharmazeutische Substanzen.
Insbesondere die Caspasen, deren Hemmung zur Verhinderung von Zelltod
führt,
oder die Familie der Secretasen, die bei der Entstehung der Alzheimerschen
Krankheit eine Rolle spielen, stellen wichtige physiologische Targets
für therapeutisch
wirksame Substanzen dar. Weiterhin sind virale Proteasen interessante
Targets für
die Therapie von Krankheiten wie AIDS oder Hepatitis.
-
Aus
den oben genannten Gründen
kommt der spezifischen Analyse und Diagnostik von Proteasen eine
besondere Bedeutung zu. Der in vivo-Nachweis von proteolytischen
Aktivitäten
erfolgt häufig über die
Detektion des entsprechenden gespaltenen Substrats der betreffenden
Protease. Hierbei haben sich insbesondere fluoreszenzbasierte Nachweisverfahren
durchgesetzt (Twining, SS; Anal Biochem 1984 Nov 15;143(1):30-4).
Diese beruhen meistens auf der Kopplung von einem Peptid, welches
die spezifische Erkennungs- und/oder Schnittstelle für die Protease
umfasst, und einem bzw. mehreren Fluoreszenzfarbstoffen. Die Farbstoffe ändern durch
die Spaltung des Peptides ihre fluoreszierenden Eigenschaften. Diese
modifizierten Fluoreszenzeigenschaften können anschließend durch
eine entsprechende Fluoreszenz-Messung nachgewiesen werden.
-
Die
einfachste Variante dieses Verfahrens ist die direkte Kopplung synthetischer
Farbstoffmoleküle
an den C- oder N-Terminus eines kurzen Substratpeptides. Der dabei
entstehende Fluoreszenz-Resonanz-Energietransfer (FRET), bzw. ein
auftretender „Selfquenching-Effekt" wird durch die Spaltung
des Peptidsubstrates aufgehoben und kann durch eine vergleichende
Fluoreszenz-Messung vor und nach der proteolytischen Spaltung des
Substratpeptides gemessen werden (zB. Jones, LJ et al.; Anal Biochem
1997 Sep 5;251(2):144-52).
-
In
einem ähnlichen
Verfahren, das ebenfalls auf dem FRET-Effekt basiert, werden in
einer Zelle fluoreszierende Fusionsproteine mit unterschiedlichen
spektralen Eigenschaften exprimiert. Die Fusionsproteine umfassen
hierbei insbesondere das „green
fluorescent protein" (GFP)
oder eine seiner Varianten und eine spezifische Substrat-Schnittstelle
für eine
Protease. Auch hier hebt die Spaltung der Substrat-Schnittstelle
einen FRET Effekt auf und erlaubt somit die Messung der proteolytischen
Aktivität
(Pollok, BA; Trends Cell Biol 1999 Feb;9(2):57-60).
-
Bei
diesen Verfahren zur Detektion Protease-abhängiger zellulärer Ereignisse
unterscheidet man zwischen solchen Verfahren, bei denen das Nachweis-Substrat
die Membran passieren kann oder nicht passieren kann. Kann das Nachweis-Substrat
die Membran nicht durchdringen, so muß das Substrat der Protease
einem Zell-Lysat bzw. einer Analyseprobe zugegeben werden. Die Aktivität wird dann
in vitro bestimmt. Bei solchen Verfahren, bei denen die Substrate
für die
Protease die Zellmembran passieren können, können die nachzuweisenden Protease-Substrate
direkt auf die Zellen gegeben. Die Protease-Substrate werden in
diesem Fall von den Zellen aufgenommen und werden in vivo in der
Zelle umgesetzt. Die proteolytische Aktivität kann somit ebenfalls in vivo – und ohne
die Zugabe von chemischen Substanzen oder künstlich synthetisierten Peptiden – gemessen
werden, was für
viele Fragestellungen einen großen
Vorteil darstellt.
-
Die
Notwendigkeit zur Bereitstellung von Protease-Substraten, die die
Zellmembran passieren können,
wird bei in vivo-Messungen durch die direkte Expression des nachzuweisenden
Protease-Substrates in den entsprechenden Zellen vermieden.
-
Die
Entdeckung des GFPs als Werkzeug in der Zellbiologie hat sich bei
der Analyse vieler intrazellulärer
Prozesse als sehr hilfreich erwiesen (Chalfie, M et al.; Science.
1994 Feb 11;263(5148):802-5). Das Grün fluoreszierende Protein der
Qualle Aequorea victoria ist 238 Aminosäuren lang. Die Wildtyp-Variante
des Proteins (GFP) absorbiert bei 395 nm und 475 nm und emittiert
bei 508 nm. Die Fluoreszenz wird verursacht durch die interne Ser-Tyr-Gly-Sequenz
an der Aminosäure-Position
65-67, die mittels Ringbildung eine p-Hydroxybenzyliden-Imidazol-Struktur
bildet.
-
Die
Aufklärung
der Kristallstruktur von GFP zeigte, dass GFP eine sehr stabile "β-barrel-Struktur" besitzt, in deren Zentrum sich der
Fluorophor befindet. Der starre Aufbau des GFP größtenteils
aus „β-Faltblattstrukturen" ist für die große Stabilität des GFPs
verantwortlich.
-
In
den vergangenen Jahren wurden durch die Einführung von Mutationen in die
Wildtyp-Form des
Proteins GFP zahlreiche Varianten des GFPs, unter anderem auch Farb-Varianten, entwickelt.
Besonders zu erwähnen
sind die Varianten mit unterschiedlichen spektralen Eigenschaften
(Farb-Varianten), wie insbesondere das YFP (Yellow Fluorescent Protein),
das CFP (Cyan Fluorescent Protein) und das BFP (Blue Fluorescent Protein),
sowie Varianten des GFPs mit unterschiedlicher Expressionstärke und
Lebensdauer, wie insbesondere EGFP (Enhanced green fluorescent protein),
EYFP (Enhanced yellow fluorescent protein), ECFP (Enhanced cyan
fluorescent protein), sowie pH-sensitive Varianten (
EP804457B ;
EP886644 ;
EP851874B ).
-
Darüber hinaus
werden GFP und seine Varianten auch als Reporter-Gene für die Detektion
von Protein-Protein-Interaktionen und durch Kopplung an entsprechende
Sensormoleküle
auch als Calcium-Indikatoren verwendet (
EP949269 ; WO9830715; WO0071565).
-
Wie
oben bereits dargelegt, können
GFPs auch zur Analyse der Aktivität von Proteasen verwendet werden.
Die hierzu bekannten Verfahren beruhen auf dem FRET-Effekt. Hierbei
werden in der Regel zwei Varianten des GFPs benutzt, bei denen sich
das Emissions- und das Absorptionsspektrum überlagern. Befinden sich beide
GFP-Varianten in großer
räumlicher
Nähe, so
erfolgt nach Anregung der Variante A (z.B. CFP) durch FRET die Anregung
der Variante B (z.B. YFP). Die Aminosäuresequenzen für beide
Varianten sind durch eine entsprechende Protease-Schnittstelle miteinander
verknüpft,
so daß ein
permanenter FRET-Effekt auftritt. Wird diese Verbindung durch Proteolyse
getrennt, so verändert
sich das Verhältnis
der Emissionsmaxima der beiden GFP-Varianten, was anschließend gemessen
werden kann (WO0073437).
-
Eine
sehr ähnliche
Methode zur Bestimmung von Proteaseaktivität mit Hilfe von GFP ist der
Bioluminescence Resonanz Energie Transfer (BRET). BRET basiert generell
auf dem selben Prinzip wie der beschriebene FRET-Sensor, die Energie
wird lediglich nicht durch die Anregung einer GFP-Variante A erzeugt,
sondern durch die Aktivierung eines chemielumineszierenden Proteins.
Die Messung der Protease-Aktivität
erfolgt hier wiederum über
die Änderung
des Emissionsmaximums von GFP.
-
Nachteil
dieser FRET-basierten Verfahren ist in erster Linie der relativ
große
instrumentelle Aufwand bei der Detektion. Darüber hinaus sind die Unterschiede
der Emissionspektra häufig
sehr schwach und machen das System sehr anfällig für Fehler.
-
Den
direkten Nachweis einer Proteaseaktivität mittels fluoreszierender
Proteine erlaubt ein Verfahren, bei dem, in das ansonsten sehr proteolysestabile
GFP, an bestimmten Stellen Protease-Schnittstellen insertiert werden.
Diese Insertionen sind so gewählt,
dass bei Proteolyse die Fluoreszenz verloren geht (Chiang, CF et al.;
Arch. Biochem. Biophys. 2001, 394, 229). Dieses Verfahren erfordert
jedoch eine sehr starke und eine über längere Zeit anhaltende Proteaseaktivität, um eine
signifikante Abnahme der Fluoreszenz des permanent nachsynthetisierten
GFPs zu detektieren. Dadurch wird die Vielfalt der analysierbaren
Proteaseaktivitäten stark
begrenzt. Insbesondere zeitlich kurz anhaltende, transiente und
schwache Proteaseaktivitäten
können mit
Hilfe dieses Verfahrens nicht detektiert und analysiert werden.
-
Ein
weiteres Verfahren, in welchem GFP zur Detektion benutzt wird, beruht
auf der Änderung
der Lokalisation eines fluoreszierenden Proteins. Dabei wird ein
Fusionsprotein exprimiert, welches GFP oder eine seiner Varianten
umfasst. Das GFP ist hierbei am N-Terminus über eine spezifische Protease-Schnittstelle
mit einem Kernexportsignal fusioniert und am C-Terminus des Fusionsproteins
befindet sich ein Kernimportsignal. Das Auftreten einer Protease-Aktivität führt zur
Abspaltung des Kernexportsignals. Dies führt zur Akkumulation der Fluoreszenz
im Kern (BD-Bioscience ApoAlert Caspase-3-sensor; Clontechniques,
2002, 4). Nachteil dieses Verfahrens ist, daß es eine entsprechend starke
Protease-Aktivität
voraussetzt, um ein eindeutig positives Signal zu erhalten. Die
Sensitivität
des Verfahrens ist somit begrenzt. Die automatische Detektion der
Lokalisationsänderung
des fluoreszierenden Proteins ist sehr aufwendig und ungenau, daher
ist das Verfahren auch nur bedingt geeignet zum Hochdurchsatz-Screening.
-
Ein
Verfahren, das den variablen Einsatz von Reportergenen erlaubt,
ist das protease based gene switching system, das in WO 99/11801
beschrieben wird. Hierbei wird ein Transkriptionsaktivator über eine spezifische
Protease-Schnittstelle an einer Transmembrandomäne verankert und somit inaktiviert.
Proteolytische Spaltung führt
zur Freisetzung des Aktivators, der anschließend die Transkription eines
zuvor inserierten Reportergens anschaltet. Ein Nachteil der Methode
ist die stabile Lokalisation des Substrates an der Zellmembran:
die Lokalisation der Protease-Schnittstelle an der Membran erfordert,
daß auch
die Protease an der Membran lokalisiert ist bzw. in deren Nähe gelangt.
Darüber
hinaus bedeutet der Nachweis der Reportergen-Expression eine in
vielen Fällen
unerwünschte
zeitliche Verzögerung
zwischen dem zu analysierenden Ereignis, d.h. dem Proteolyse-abhängigen Ereignis
in der Zelle, und dem Detektionssignal.
-
Die
oben genannten Verfahren zur Detektion von Protease-Aktivitäten beinhalten
jeweils mindestens einen der folgenden Nachteile:
- – keine
Detektion in vivo
- – relativ
großer
instrumenteller Aufwand bei der Detektion im Rahmen der FRET-basierten Verfahren
- – geringe
Sensitivität
des Verfahrens
- – große Fehleranfälligkeit
des Verfahrens
- – zeitliche
Verzögerung
zwischen dem zu analysierenden Ereignis, d.h. dem Proteolyse-abhängigen Ereignis
in der Zelle, und dem Detektionssignal
- – geringe
Eignung zum Hochdurchsatz-Screening.
-
Es
ist somit Aufgabe der vorliegenden Erfindung, ein Verfahren zur
Detektion von Protease-Aktivitäten bzw.
von Protease-abhängigen
Ereignissen in der Zelle bereitzustellen, welches zumindest einige
der obigen Nachteile der Verfahren nach dem Stand der Technik überwindet.
Weiterhin ist es Aufgabe der Erfindung, Nukleinsäuren und Proteine zur Durchführung dieses
Verfahrens bereitzustellen.
-
Diese
Aufgabe wird zum einen gelöst
durch die Bereitstellung eines Verfahrens, welches die Detektion eines
fluoreszierenden Sensorproteins in Abhängigkeit von der Aktivität einer
bestimmten Protease in vivo erlaubt. Grundlage des Systems ist die
heterologe Expression einer nicht fluoreszierenden Variante eines
in seiner Wildtyp-Version fluoreszierenden Proteins, wie insbesondere
die heterologe Expression einer Variante der GFP-Familie, welche
eine kurze, von Protease-Schnittstellen ein- oder beidseitig flankierte
Domäne
im N-terminalen Bereich des Proteins umfasst. Diese Domäne weist
vorzugsweise eine α-helikale
räumliche
Struktur auf. Die von Protease-Schnittstellen ein- oder beidseitig
flankierte, vorzugsweise α-helikale
Stör-Domäne befindet
sich hierbei entweder am unmittelbaren N-Terminus des Proteins oder
am N-Terminus eines seiner N-terminal trunkierten Fragmente oder
ist nach der Aminosäure
mit der Position 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14,
15, vorzugsweise aber nach der Aminosäure mit der Position 2, 3,
4, 5, 6, 7, 8, 9, gezählt
vom N-Terminus des Proteins, ins Protein insertiert.
-
Durch
die Anwesenheit der ein- oder beidseitig von Protease-Schnittstellen
flankierten Stör-Domäne am unmittelbaren
N-Terminus (Anhängen
der Stör-Domäne) oder
im Bereich des N-Terminus (Insertion der Stör-Domäne) verliert das Protein seine
fluoreszierenden Eigenschaften bzw. wird seine Fluoreszenz derart
in ihrer Intensität
reduziert, dass Zellen, die dieses Protein exprimieren, mit Standardverfahren
nicht mehr von solchen Zellen unterscheidbar sind, die dieses Protein
nicht exprimieren. Das so modifizierte Protein erscheint somit – im Vergleich
zum Wildtyp-Protein wie ein nicht fluoreszierendes Protein.
-
Nach
der proteolytischen Entfernung der Stör-Domäne durch eine in der Zelle
auftretende, zu detektierende Protease-Aktivität kommt es jedoch wieder zu
einer drastischen Erhöhung
der Fluoreszenz-Intensität bzw.
erlangt das Protein seine fluoreszierenden Eigenschaften zurück. Die
Fluoreszenz des Proteins kann somit in Abhängigkeit von dem Auftreten
einer Protease-Aktivität
in der betreffenden Zelle detektiert werden.
-
Dieses
Verfahren erlaubt den unmittelbaren Nachweis einer proteolytischen
Aktivität
in lebendigen Zellen, Geweben oder Organismen in vivo. Weiterhin
erlaubt ein solches Verfahren durch das Protease-abhängige „Umschalten" eines nicht fluoreszierenden
Proteins in ein fluoreszierendes Protein eine relativ einfache Detektion
des Fluoreszenz-Signals
vor einem niedrigen Hintergrund-Rauschen, insbesondere im Vergleich
zu der recht aufwendig zu messenden Fluoreszenzänderung bei den FRET-basierten
Verfahren. Dies begründet
die hohe Sensitivität
und geringe Fehleranfälligkeit
des Verfahrens, sowie dessen Eignung zum Hochdurchsatz-Screening.
Ein weiterer Vorteil des Verfahrens ist, daß es zwischen der Protease-Aktivität in der
Zelle, d.h. zwischen dem zu analysierenden Ereignis und dem Detektionssignal
zu keinerlei zeitlichen Verzögerung
kommt. Da auch eine geringe oder transiente, d.h. zeitlich begrenzte
Protease-Aktivität
in der Zelle zu einem fluoreszierenden Protein in der Zelle führt, eignet
sich das erfindungsgemäße Verfahren
auch zur Detektion transienter Protease-abhängiger Ereignisse, was von
großem
Vorteil ist. Weiterhin kann das nicht fluoreszierende, aber proteolytisch
zur Fluoreszenz aktivierbare Protein an beliebigen Stellen in und
außerhalb der
Zelle lokalisiert werden, z.B. durch nukleäre Import- und Exportsignale,
sowie durch membranständige
Domänen
oder Präsequenzen
für sekretorische
Proteine. Die Fluoreszenzdetektion dieser Sensorproteine ist damit
nicht auf ein bestimmtes Kompartiment der Zelle beschränkt.
-
Die
erfindungsgemäße Aufgabe
wird zum anderen auch durch die Bereitstellung solcher Nukleinsäuren und
Proteine gelöst,
die zur Durchführung
des erfindungsgemäßen Verfahrens
notwendig sind.
-
So
wird die Aufgabe insbesondere durch die Bereitstellung einer Nukleinsäure gelöst, die
für ein
nicht fluoreszierendes, aber proteolytisch zur Fluoreszenz aktivierbares
Protein kodiert, welches an seinem N-Terminus mindestens eine ein-
oder beidseitig von Protease-Schnittstellen
flankierte Stör-Aminosäuresequenz umfasst.
-
Vorzugsweise
umfasst hierbei das nicht fluoreszierende, aber proteolytisch zur
Fluoreszenz aktivierbare Protein eine Sequenz nach SEQ ID No. 1
oder nach SEQ ID No. 2 oder eines ihrer Fragmente mit mindestens
5 Aminosäuren
Länge,
an deren N-Terminus die ein- oder beidseitig von Protease-Schnittstellen
flankierte Stör-Aminosäuresequenz
fusioniert ist.
-
Besonders
bevorzugt wird hierbei als nicht fluoreszierendes, aber proteolytisch
zur Fluoreszenz aktivierbares Protein eine natürliche oder artifizielle Variante
der GFP-Familie
aus Aequorea victoria, insbesondere EGFP (Acc. No. U76561), EYFP
(Acc. No. AJ510163, kodiert von der Region 6103-6822), ECFP (Acc.
No. AJ510158, kodiert von der Region 6058-6780), GFP (Acc. No. X83959
), YFP (Acc. No. AY189981, kodiert von der Region 1603-2331) oder
CFP (Acc. No. BD136947) eingesetzt, wobei diese Variante der GFP-Familie
an ihrem N-Terminus eine ein- oder beidseitig von Protease-Schnittstellen flankierte
Stör-Aminosäuresequenz umfasst.
-
Alternativ
wird die Aufgabe insbesondere durch die Bereitstellung einer Nukleinsäure gelöst, die
für ein nicht
fluoreszierendes, aber proteolytisch zur Fluoreszenz aktivierbares
Protein kodiert, welches mindestens eine ein- oder beidseitig von
Protease-Schnittstellen flankierte Stör-Aminosäuresequenz umfasst, die nach
der Aminosäure
mit der Position 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14,
15, vorzugsweise aber nach der Aminosäure mit der Position 2, 3,
4, 5, 6, 7, 8, 9, gezählt
vom N-Terminus des Proteins, im Protein insertiert ist.
-
In
einer bevorzugten Ausführungsform
kodiert die erfindungsgemäße Nukleinsäure für ein nicht
fluoreszierendes, aber proteolytisch zur Fluoreszenz aktivierbares
Protein, welches an seinem N-Terminus eine Aminosäuresequenz
nach Sequenz nach SEQ ID No. 1 umfasst und bei dem mindestens eine
ein- oder beidseitig von Protease-Schnittstellen flankierte Stör-Aminosäuresequenz
nach der Aminosäure
mit der Position 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14 oder
15, gezählt
vom N-Terminus des Proteins, insertiert ist.
-
In
einer weiteren bevorzugten Ausführungsform
kodiert die erfindungsgemäße Nukleinsäure für ein nicht
fluoreszierendes, aber proteolytisch zur Fluoreszenz aktivierbares
Protein, welches an seinem N-Terminus eine Aminosäuresequenz
nach Sequenz nach SEQ ID No. 2 umfasst und bei dem mindestens eine
ein- oder beidseitig von Protease-Schnittstellen flankierte Stör-Aminosäuresequenz
nach der Aminosäure
mit der Position 2, 3, 4, 5, 6, 7, 8 oder 9, gezählt vom N-Terminus des Proteins,
insertiert ist.
-
Besonders
bevorzugt sind solche Nukleinsäuren,
die für
ein nicht fluoreszierendes, aber proteolytisch zur Fluoreszenz aktivierbares
Protein kodieren, wobei dieses Protein eine natürliche oder artifizielle Variante der
GFP-Familie aus Aequorea victoria ist, welche mindestens eine ein-
oder beidseitig von Protease-Schnittstellen flankierte Stör-Aminosäuresequenz
umfasst, die nach der Aminosäure
mit der Position 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14 oder
15, vorzugsweise nach der Aminosäure
mit der Position 2, 3, 4, 5, 6, 7, 8 oder 9, gezählt vom N-Terminus des Proteins,
in die Variante der GFP-Familie insertiert ist. Die natürlichen
oder artifiziellen Varianten der GFP-Familie aus Aequorea victoria
sind insbesondere die GFP-Varianten: EGFP (Acc. No. U76561), EYFP
(Acc. No. AJ510163, kodiert von der Region 6103-6822), ECFP (Acc.
No. AJ510158, kodiert von der Region 6058-6780), GFP (Acc. No. X83959
), YFP (Acc. No. AY189981, kodiert von der Region 1603-2331) oder
CFP (Acc. No. BD136947).
-
Die
Stör-Aminosäuresequenz
kann generell jede beliebige Länge
größer als
3 Aminosäure,
vorzugsweise größer als
5 Aminosäuren,
stärker
bevorzugt als 7 Aminosäuren,
insbesondere größer als
10 Aminosäuren
besitzen. In einer besonders bevorzugten Ausführungsform besitzt die Stör-Aminosäuresequenz
eine Länge
zwischen 5 und 100 Aminosäuren.
-
Die
Stör-Aminosäuresequenz
kann generell jede beliebige Aminosäuresequenz besitzen. Vorzugsweise
besitzt die Stör-Aminosäuresequenz
aber eine Aminosäuresequenz,
die dazu neigt, α-Helix-Strukturen auszubilden.
Um diese Voraussetzungen zu erfüllen,
umfasst die Stör-Aminosäuresequenz
vorzugsweise solche Aminosäuren,
die „Helixbildner" sind, wie insbesondere
Leucin und Methionin, sowie polare oder geladene Aminosäuren wie
insbesondere Glutamat, Aspartat, Arginin und Lysin. Letztere sollen
die Gesamthydrophobizität
senken und das Entstehen einer Transmembrandomäne verhindern.
-
Insbesondere
kann die Stör-Aminosäuresequenz
eine Aminosäuresequenz
nach SEQ ID No. 3 oder ein mindestens 5 Aminosäuren langes Fragment dieser
Aminosäuresequenz
nach SEQ ID No. 3 besitzen.
-
Die
Stör-Aminosäuresequenzen
sind in den erfindungsgemäßen nicht
fluoreszierenden, aber proteolytisch zur Fluoreszenz aktivierbaren
Proteinen ein- oder beidseitig von Protease-Schnittstellen flankiert.
-
Als „Protease-Schnittstelle" wird eine kurze
Aminosäuresequenz
definiert, die von einer spezifischen Protease erkannt und spezifisch
geschnitten wird. So wird beispielsweise die Protease-Schnittstelle „ENLYFQG" spezifisch von der
NIa-Protease des Tobacco Etch Virus (im folgenden „TEV-Protease" oder „TEV" genannt) erkannt
und zwischen Q und G spezifisch von der TEV-Protease geschnitten.
Andere Proteasen besitzen hingegen andere spezifische Protease-Schnittstellen,
die größtenteils
dem Fachmann bekannt sind.
-
Im
Rahmen der vorliegenden Erfindung können als Protease-Schnittstellen
generell alle bekannten spezifischen Protease-Schnittstellen eingesetzt
werden, die dem Fachmann bekannt sind. Man wird jeweils die Protease-Schnittstelle
zur ein- oder beidseitigen Flankierung der Stör-Aminosäuresequenz einsetzen, deren zugehörige Protease-Aktivität im Rahmen
des Verfahrens detektiert oder charakterisiert werden soll. Als
Protease-Schnittstelle
können
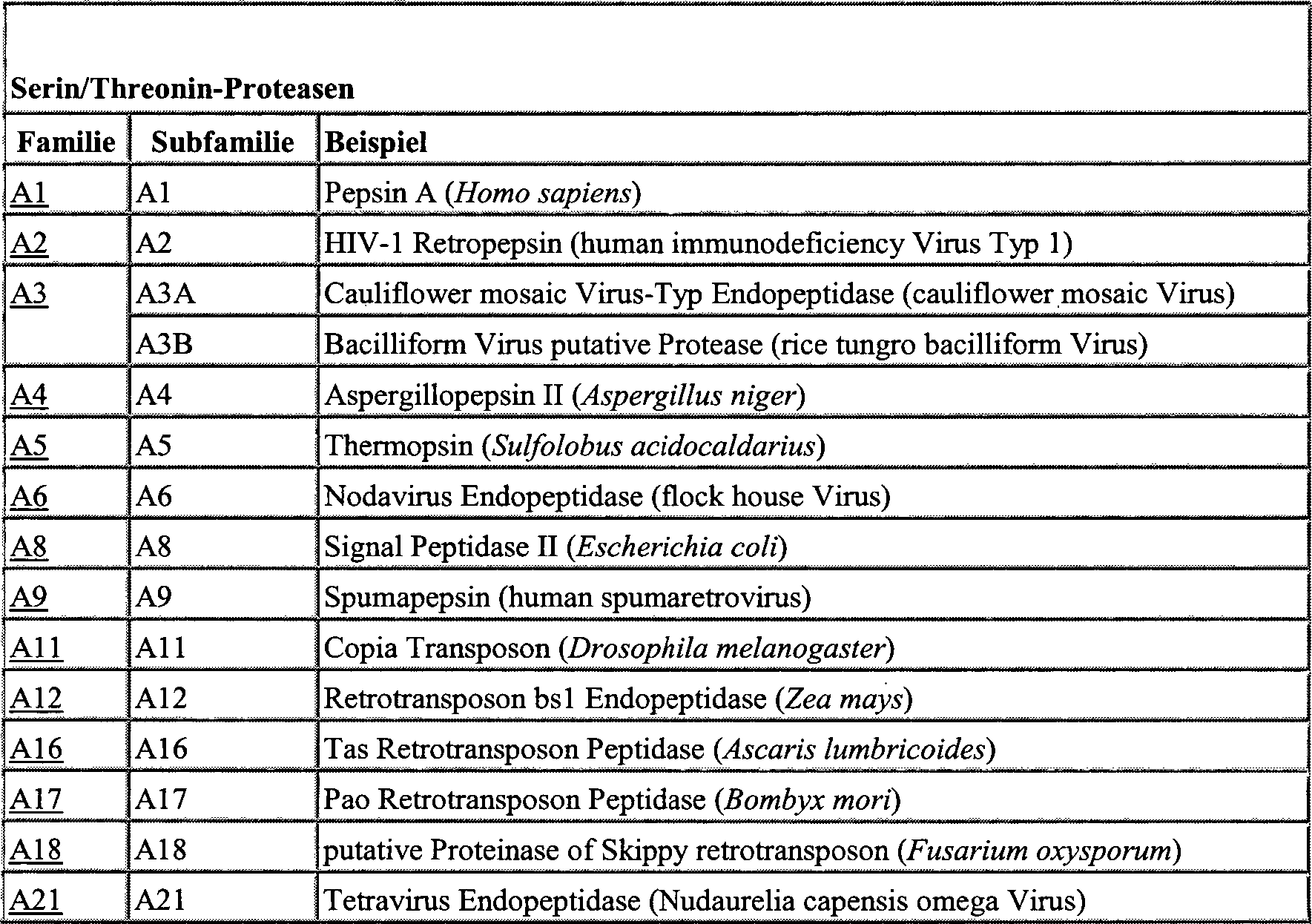
vorzugsweise die spezifischen Schnittstellen der Serin-/Threonin-Proteasen, der Cystein-Proteasen,
der Aspartat-Proteasen, der Metalloproteasen und der unklassifizierten
Proteasen eingesetzt werden. Insbesondere können die Erkennungs- und Schnittstellen
der Proteasen aus Tabelle 1 als Protease-Schnittstellen zur Flankierung
der Stör-Aminosäuresequenz
verwendet werden. Viele der entsprechenden Schnittstellen der Proteasen
aus Tabelle 1 sind dem Fachmann aus der Fachliteratur bekannt.
-
-
-
-
-
-
-
-
Ein
weiterer Gegenstand der Erfindung sind solche Nukleinsäuren, die
für ein
Fusionsprotein eines der oben genannten nicht fluoreszierenden,
aber proteolytisch zur Fluoreszenz aktivierbaren Proteine kodieren. Ein
solches Fusionsprotein umfasst ein nicht fluoreszierendes, aber
proteolytisch zur Fluoreszenz aktivierbares Protein, welches mindestens
eine ein- oder beidseitig von Protease-Schnittstellen flankierte
Stör-Aminosäuresequenz
entweder am N-Terminus angehängt
oder als Insertion nach der Aminosäure mit der Position 1, 2,
3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15 enthält, fusioniert mit einem weiteren
Protein oder mit einer weiteren Proteindomäne. Das weitere Protein oder
die weitere Proteindomäne
kann hierbei in der Regel sowohl am N-Terminus als auch am C-Terminus
des Fusionsproteins lokalisiert sein. Die Fusion am C-Terminus ist
lediglich insoweit bevorzugt, als daß eine am N-Terminus lokalisierte
weitere Proteindomäne
durch die Protease-Aktivität,
die die von Protease-Schnittstellen flankierte Stör-Aminosäuresequenz
am N-Terminus abspaltet, auch zur Abspaltung der weiteren Proteindomäne führen würde.
-
„Weitere
Proteindomänen" im Sinne der obigen
Definition des Fusionsproteins können
vorzugsweise verschiedene Lokalisationsdomänen sein, die für die Kompartimentierung
des Fusionsproteins in der Zelle verantwortlich sind, wie insbesondere
ein Kernlokalisationssignal („nuclear
localization signal" NLS),
ein Kernexportsignal („nuclear
export signal" NES),
eine Membrandomäne,
enthaltend vorwiegend hydrophobe Aminosäuren, eine klassische Präsequenz,
die das Protein als „sekretorisches
Protein" kennzeichnet,
sowie weitere bekannte Lokalisationssignale für verschiedene Zellkompartimente.
-
Ein
weiterer Gegenstand der Erfindung ist eine Expressionskassette,
die eine der oben genannten Nukleinsäuren bzw. Nukleinsäuresequenzen
unter der Kontrolle eines Promoters umfassen.
-
Der
Promoter kann hierbei jeder bekannte Promoter sein, der in der Wirtszelle,
in die die Expressionskassette eingebracht werden soll, aktiv ist,
d.h. in dieser Wirtszelle die Transkription des nachgeschalteten
Reportergens aktiviert. Der Promoter kann hierbei ein konstitutiver
Promoter sein, der das nachgeschaltete Reportergen ständig exprimiert,
oder ein nicht-konstitutiver Promoter, der nur zu definierten Zeitpunkten
im Laufe der Entwicklung oder unter bestimmten Umständen (insbesondere
unter Einfluß eines
Transkriptionsaktivators oder in Abwesenheit eines Transkriptionsrepressors)
exprimiert. Erfindungsgemäße Expressionskassetten, die
als Promoter beispielsweise den CMV-Promoter enthalten, eignen sich für die Expression
des nachgeschalteten nicht fluoreszierenden, aber proteolytisch
zur Fluoreszenz aktivierbaren Reporter-Proteins in eukaryontischen,
insbesondere in Säugetier-Wirtszellen
und Hefe-Wirtszellen. Erfindungsgemäße Expressionskassetten, die
als Promoter beispielsweise den lac-Promoter enthalten, eignen sich
für die
Expression des nachgeschalteten nicht fluoreszierenden, aber proteolytisch
zur Fluoreszenz aktivierbaren Reporter-Proteins in prokaryontischen,
insbesondere in Bakterien-Wirtszellen.
-
Der
Ausdruck „unter
der Kontrolle" eines
Promoters bedeutet, daß die
Promotersequenz und die Sequenz kodierend für das zu exprimierende nicht
fluoreszierende, aber proteolytisch zur Fluoreszenz aktivierbare
Reporter-Protein so miteinander verknüpft sind, daß die Expression
des Reporter-Proteins möglich
ist.
-
Die
erfindungsgemäße Expressionskassette
kann gegebenenfalls weitere Kontrollsequenzen enthalten. Unter einer
Kontrollsequenz wird eine beliebige Nukleotidsequenz verstanden,
die die Expression des nicht fluoreszierenden, aber proteolytisch
zur Fluoreszenz aktivierbaren Reporter-Proteins beeinflußt, wie
insbesondere der Promoter, eine Operatorsequenz, d.h. die DNA-Bindungstelle
für einen
Transkriptionsaktivator oder einen Transkriptionsrepressor, eine
Terminator-Sequenz, eine Polyadenylierungssequenz oder eine Ribosombindungsstelle.
-
Ein
weiterer Gegenstand der Erfindung ist ein rekombinanter Vektor,
der eine der obigen erfindungsgemäßen Expressionskassetten enthaltend
ein nicht fluoreszierendes, aber proteolytisch zur Fluoreszenz aktivierbares
Protein umfasst.
-
Ein
solcher rekombinanter Vektor kann zusätzlich eine Nukleotidsequenz
enthalten, durch die der Vektor sich in der betreffenden Wirtszelle
replizieren kann. Solche Nukleotidsequenzen werden in der Regel „origin of
replication" (deut.
Replikationsursprung) genannt. Beispiele für solche Nukleotidsequenzen
sind der SV40-Replikationsursprung,
der in Säugetier-Wirtszellen
zum Einsatz kommt, und in Hefe-Wirtszellen
die Hefe-Plasmid 2μ Replikationsgene
REP 1-3.
-
Der
rekombinante Vektor kann weiterhin einen oder mehrere Selektionsmarker
enthalten. Unter einem Selektionsmarker versteht man ein Gen, welches
unter der Kontrolle eines Promoters steht und welches für ein Protein
kodiert, das einen physiologischen Defekt der Wirtszelle komplementiert.
Selektionsmarker stellen insbesondere das Gen kodierend für die Dihydrofolat
Reduktase (DHFR) dar, oder auch ein Gen, welches die Resistenz gegen
Antibiotika, wie insbesondere Ampicillin, Kanamycin, Tetracyclin,
Blasticidin, Gentamycin, Chloramphenicol, Neomycin oder Hygromycin
bewirkt.
-
Eine
große
Anzahl von rekombinanten Vektoren zur Expression eines Zielproteins
in prokaryontischen oder eukaryontischen Wirtszellen sind nach dem
Stand der Technik bekannt und viele sind auch kommerziell erhältlich.
-
Ein
weiterer Gegenstand der Erfindung ist eine Wirtszelle, die mit dem
erfindungsgemäßen rekombinanten
Vektor transient oder stabil transformiert worden ist.
-
Die
Auswahl der geeigneten Wirtszelle, hängt von einer großen Anzahl
an Faktoren ab, die dem Fachmann bekannt sind. Diese Faktoren beinhalten
insbesondere den gewählten
Vektortyp, die Toxizität
des exprimierten Proteins für
die betreffende Wirtszelle, die zu beantwortende Fragestellung,
die Expressionscharakteristika und physiologischen Wechselwirkungen
des betreffenden Zielproteins in der Wirtszelle, die Sicherheitsrisiken
und Kosten. Generell kann jede beliebige pro- oder eukaryontische
Zelle bzw. Organismus als Wirtszelle eingesetzt werden.
-
Beispiele
für geeignete
prokaryontische Wirtszellen sind gram-positive Bakterien wie insbesondere Bacillus
subtilis, Bacillus licheniformis, Bacillus brevis, Streptomyces
lividans etc. oder gram-negative Bakterien wie insbesondere E. coli.
-
Beispiele
für geeignete
eukaryontische Wirtszellen sind die Spezies der Saccharomyces oder
Schizosaccharomyces, insbesondere Saccharomyces cerevisae.
-
Beispiele
für Zelllinien,
die von Säugetieren
stammen und die ebenfalls als Wirtszellen in Frage kommen, sind
insbesondere die Zelllinien COS-1 (ATCC CRL 1650), COS-7 (ATCC CRL
1651), CHO K1 (ATCC CCL 61), NIH 3T3 (ATCC CRL 1658), HeLa (ATCCL
2), MRC-5 (ATCC CCL 171), HEK 293 (ATCC CRL1573).
-
Der
rekombinante Vektor kann in die betreffende Wirtszelle durch jegliche
Transfektions-, Transformations- oder Injektionstechnik, die dem
Fachmann bekannt ist, eingeführt
werden. Insbesondere kann der rekombinante Vektor durch eine der
folgenden Techniken in die betreffende Wirtszelle eingeführt werden:
Calciumphosphat-Präzipitation,
Elektroporation, Protoplastenfusion, Nukleinsäure-Injektion, Lipofektion, „gene gun" unterstützte Techniken,
Infektion mit Virus-Partikeln oder Virus-abgeleiteten Partikeln
und Protein-Transduktion mit TAT oder TAT-ähnlichen Sequenzen.
-
Die
Wirtszelle kann hierbei entweder transient oder stabil transformiert
worden sein. Bei der „transienten
Transformation" einer
Zelle bleibt der eingeführte
Vektor in der Zelle in der Regel autonom, d.h. er integriert sich
nicht in das Wirtszellgenom. Bei Teilung der transient transformierten
Zellen in Tochterzellen wird der Vektor nicht mit übertragen.
Dies führt
dazu, daß der
eingeführte
Vektor sich nach mehreren Wachstumscyclen der transformierten Zellen
so lange „herausverdünnt" bis die meisten
Zellen keinen Vektor mehr enthalten. Auch bei transient transformierten
Zellen kann man den Verlust des Vektors nach mehreren Wachstumscyclen
unterbinden, indem der Selektionsdruck auf die Gegenwart des rekombinanten
Vektors, der einen Selektionsmarker wie oben definiert umfasst,
aufrecht erhalten wird.
-
Bei
der „stabilen
Transformation" einer
Zelle integriert sich der eingeführte
Vektor, der meist in linearisierter Form eingeführt wird, in das Wirtsgenom
der Zelle. Bei Teilung der stabil transformierten Zellen in Tochterzellen
werden demnach die ursprünglichen
Vektorsequenzen als Bestandteil des Wirtsgenoms mit übertragen.
Eine auf einem rekombinanten Vektor befindliche Expressionskassette
wird daher dauerhaft, d.h. über eine
große
Anzahl von Wachstumscyclen hinweg, in den Tochterzellen exprimiert.
-
Transiente,
sowie stabile Transformationstechniken sind dem Fachmann bekannt
und in gängigen Nachschlagewerken
nachzulesen (Freshney, IR; Culture of Animal Cells, 2000, 4th Ed.
Wiley-Liss).
-
Ein
weiterer Gegenstand der Erfindung ist ein Kit zur Detektion und/oder
zur Analyse von von Protease-Aktivitäten oder von Protease-abhängigen Ereignissen,
welcher mindestens eine der folgenden Komponenten umfasst:
- a) eine Nukleinsäure kodierend für ein nicht
fluoreszierendes, aber proteolytisch zur Fluoreszenz aktivierbares
Reporter-Protein, welches mindestens eine ein- oder beidseitig von
Protease-Schnittstellen flankierte Stör-Aminosäuresequenz entweder am N-Terminus
angehängt
oder als Insertion nach der Aminosäure mit der Position 1, 2,
3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15 enthält, oder eine Nukleinsäure kodierend
für ein Fusionsprotein
dieses Reporter-Proteins,
- b) eine Expressionskassette umfassend eine Nukleinsäure nach
a) unter der Kontrolle eines Promoters,
- c) einen rekombinanten Vektor, der mindestens eine der Expressionskassetten
nach b) umfasst,
- d) eine Wirtszelle, die mit mindestens einem rekombinanten Vektor
nach c) transient oder stabil transformiert worden ist,
- e) ein Protein, welches von einer Nukleinsäure nach a), von einer Expressionskassette
nach b) oder von einem rekombinanten Vektor nach c) kodiert wird
oder welches von einer Wirtszelle nach d) exprimiert wird.
-
Die
Detektion und/oder Analyse von Protease-Aktivitäten oder von Protease-abhängigen Ereignissen erfolgt
vorzugsweise in vitro in dem Zellextrakt einer Zelle, die mit einem
erfindungsgemäßen rekombinanten Vektor
transformiert worden ist, insbesondere aber in vivo in einer Zelle,
die mit einem erfindungsgemäßen rekombinanten
Vektor transformiert worden ist.
-
Die
Detektion und/oder Analyse von Protease-Aktivitäten oder von Protease-abhängigen Ereignissen erfolgt
vorzugsweise mittels Fluoreszenzmikroskopie von Zellen, die mit
einem erfindungsgemäßen rekombinanten
Vektor transformiert worden sind. Die Detektion und/oder Analyse
von Protease-Aktivitäten
oder von Protease-abhängigen
Ereignissen kann weiterhin mittels Fluoreszenzspekroskopie bzw.
mittels Fluorescence Aided Cell Sorting („FACS") erfolgen.
-
Mit
Hilfe des erfindungsgemäßen Verfahrens
bzw. mit Hilfe des erfindungsgemäßen Kits
können
beliebige Protease-Aktivitäten
in Zellen, Zellextrakten, Zellüberständen, in
Fraktionen von Zellextrakten oder Zellüberständen detektiert und analysiert
werden.
-
Insbesondere
können
solche Protease-Aktivitäten
detektiert und analysiert werden, die in der Zelle lediglich transient,
d.h. vorübergehend
und nur in kurzen Zeiträumen
auftreten. Da auch eine nur kurzzeitig auftretende Protease-Aktivität in der
Zelle zur proteolytischen Entfernung der Stör-Aminosäuresequenzen aus dem Reporter-Protein – und damit
letztlich zum Umschalten von einem nicht fluoreszierenden Reporter
in einen fluoreszierenden Reporter – führt, können mit dem erfindungsgemäßen Verfahren
auch vorzugsweise transient auftretende, sowie schwache Protease-Aktivitäten gemessen
werden.
-
Verschiedenste
physiologisch signifikante zelluläre Ereignisse bzw. Signaltransduktionswege
beinhalten das Auftreten von Protease-Aktivitäten, insbesondere das Auftreten
von transienten, d.h. kurzzeitigen Protease-Aktivitäten in der
Zelle. Insbesondere die Apoptose ist ein solches physiologische
relevantes zelluläres Ereignis,
welches mit dem Auftreten von Protease-Aktivitäten, insbesondere mit der Aktivität der Caspasen und
der Calpaine einhergeht. Durch die Detektion der Aktivität dieser
Apoptoserelevanten Proteasen könnten daher
insbesondere apoptotische Zellen mittels des erfindungsgemäßen Verfahrens
als fluoreszierende Zellen detektiert werden.
-
Generell
können
jedoch mit den erfindungsgemäßen Kits
und/oder Verfahren alle beliebigen Protease-abhängigen Ereignisse in der Zelle
detektiert werden.
-
Weiterhin
können
mit den erfindungsgemäßen Kits
und/oder Verfahren nicht nur Protease-Aktivitäten oder Protease-abhängige Ereignisse
selbst, sondern auch solche zellulären Ereignisse detektiert und
analysiert werden, die die Ursache für das Auftreten der Protease-Aktivität in der
Zelle oder eine Folge des Auftretens der Protease-Aktivität in der
Zelle sind. Die Ursache für
das Auftreten einer bestimmten Protease-Aktivität in der Zelle kann insbesondere
auch das Auftreten einer spezifischen Protein-Protein-Interaktion
sein.
-
Ein
weiterer Gegenstand der Erfindung ist ein nicht fluoreszierendes,
aber proteolytisch zur Fluoreszenz aktivierbares Protein oder Fusionsprotein,
welches von einer der oben definierten Nukleinsäuren kodiert wird.
-
Besonders
bevorzugt sind solche Reporter-Proteine oder Reporter-Fusionsproteine,
die ein Derivat einer natürlichen
oder artifiziellen Variante der GFP-Familie aus Aequorea victoria,
insbesondere ein Derivat der Varianten EGFP (Acc. No. U76561), EYFP
(Acc. No. AJ510163, kodiert von der Region 6103-6822), ECFP (Acc.
No. AJ510158, kodiert von der Region 6058-6780), GFP (Acc. No. X83959
), YFP (Acc. No. AY189981, kodiert von der Region 1603-2331) oder
CFP (Acc. No. BD136947) sind.
-
Die
Erfindung betrifft weiterhin ein Verfahren zur Detektion und/oder
zur Charakterisierung einer Protease-Aktivität in einer Zelle, welche die
folgenden Verfahrensschritte umfasst:
- a) Transfektion
einer Zelle
mit dem oben genannten rekombinanten Vektor zur
rekombinanten Expression eines nicht fluoreszierenden Proteins oder
Fusionsproteins, welches mindestens eine ein- oder beidseitig von
Protease-Schnittstellen flankierte Stör-Aminosäuresequenz am N- Terminus angehängt oder
durch Insertion in die oben genannten Protein-Positionen umfasst,
- b) Aktivierung einer bislang inaktiven Protease in der Zelle
oder Aktivierung der Expression einer Protease in der Zelle, wobei
die in Schritt a) genannten Protease-Schnittstellen Substrate dieser Protease
darstellen,
- c) Generierung eines fluoreszierenden Proteins oder Fusionsproteins
durch proteolytische Entfernung der mindestens einen ein- oder beidseitig
von Protease-Schnittstellen flankierten Stör-Aminosäuresequenz aus Schritt a) aus
dem nicht fluoreszierenden Protein oder Fusionsprotein aus a),
- d) Detektion der Fluoreszenz des fluoreszierenden Proteins oder
Fusionsproteins aus c).
-
In
Schritt a) wird eine geeignete Wirtszelle mit einem erfindungsgemäßen rekombinanten
Vektor zur rekombinanten Expression eines nicht fluoreszierenden
Proteins, aber proteolytisch zur Fluoreszenz aktivierbaren Reporter-Proteins
entweder transient oder stabil transformiert. Das nicht fluoreszierende
Protein besitzt hierbei entweder mindestens eine ein- oder beidseitig
von Protease-Schnittstellen flankierte Stör-Aminosäuresequenz, welche unmittelbar
am N-Terminus des Proteins angehängt
ist, oder mindestens eine ein- oder
beidseitig von Protease-Schnittstellen flankierte Stör-Aminosäuresequenz,
welche nach einer Aminosäure
mit der Position 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14,
15 im Protein insertiert ist. Das nicht fluoreszierende Protein kann
auch an seinem N-Terminus oder vorzugsweise an seinem C-Terminus
mit einer weiteren Proteindomäne fusioniert
sein. Die Transformation kann über
alle dem Fachmann bekannten Transformations- und Transfektionstechniken
erfolgen, so insbesondere durch Calciumphosphat-Präzipitation,
Elektroporation, Protoplastenfusion, Nukleinsäure-Injektion, Lipofektion, „gene gun" unterstützte Techniken,
Infektion mit Virus-Partikeln oder mit Virus-abgeleiteten Partikeln
und Protein-Transduktion mit TAT oder TAT-ähnlichen Sequenzen. Der rekombinante
Vektor besitzt hierbei vorzugsweise die oben bereits erläuterten
Eigenschaften.
-
In
Schritt b) erfolgt gegebenenfalls die Aktivierung einer bislang
inaktiven Protease in der Zelle oder auch die Aktivierung der Expression
einer Protease in der Zelle. Die in Schritt a) genannten Protease-Schnittstellen
stellen hierbei Substrate dieser aktivierten oder exprimierten Protease
aus Schritt b) dar.
-
Die
Aktivierung einer bislang inaktiven Protease in der Zelle oder die
Aktivierung der Expression einer Protease in der Zelle erfolgt vorzugsweise
dadurch, daß die
transformierte Zelle aus Schritt a) solchen Bedingungen ausgesetzt
wird, unter denen die zu detektierende Protease-Aktivität in der
Zelle oder das zu detektierende Protease-abhängige Ereignis in der Zelle
auftreten kann. Solche Bedingungen umfassen insbesondere die Expression
oder Aktivierung einer weiteren Komponente (insbesondere eines weiteren
Proteins) in der Zelle, welche eventuell unmittelbar oder mittelbar
zu der betreffenden Protease-Aktivität führen kann, die Zugabe von Substanzen
zur unmittelbaren oder mittelbaren Inhibierung oder Aktivierung
der betreffenden Protease-Aktivität, sowie das Bereitstellen
bestimmter physikalisch-chemischer Bedingungen, wie beispielsweise Temperaturänderungen,
pH-Änderungen
etc., die unmittelbar oder mittelbar zu der betreffenden Protease-Aktivität führen können.
-
Das
erfindungsgemäße Verfahren
wird vorzugsweise zur Detektion und zur Charakterisierung von Apoptose-relevanten
Protease-Aktivitäten
eingesetzt. Hierbei könnten
die transformierten Zellen aus Schritt a) im Rahmen von Schritt
b) solchen Bedingungen ausgesetzt werden, die bekanntermaßen Apoptose
verursachen.
-
Das
erfindungsgemäße Verfahren
wird vorzugsweise zur Detektion und zur Charakterisierung von solchen
Protease-Aktivitäten
eingesetzt werden, die infolge einer spezifischen Protein-Protein-Interaktion
in der betreffenden Zelle auftreten. Hierbei könnten die transformierten Zellen
aus Schritt a) im Rahmen von Schritt b) mit einem oder mit zwei
Expressionsvektoren transformiert werden, die für zwei verschiedene, eventuell
interagierende Fusionsproteine kodieren.
-
In
Schritt c) wird die mindestens eine ein- oder beidseitig von Protease-Schnittstellen
flankierten Stör-Aminosäuresequenz
aus Schritt a) aus dem nicht fluoreszierenden Protein oder Fusionsprotein
aus Schritt a) in Abhängigkeit
von dem Auftreten einer Protease-Aktivität in Schritt
b) proteolytisch entfernt. Eine erfolgreiche proteolytische Entfernung
der Stör-Aminosäuresequenz
im zunächst
nicht fluoreszierenden Reporter-Protein oder Fusionsprotein führt dazu,
daß ein
fluoreszierendes Reporter-Protein oder Fusionsprotein generiert
wird, dessen Fluoreszenz detektierbar ist.
-
In
Schritt d) wird die in Abhängigkeit
von der Protease-Aktivität
auftretende Fluoreszenz des Reporter-Proteins oder des Reporter-Fusionsproteins
mittels geeigneter Meßmethoden
detektiert. Im allgemeinen eignen sich zur Detektion des Fluoreszenz-Signals
alle dem Fachmann bekannten Verfahren, die die folgenden Schritte
beinhalten:
- - Anregung mit Licht der Absorptionswellenlänge des
fluoreszierenden Proteins
- - Detektion von Licht mit der Emissionswellenlänge des
fluoreszierenden Proteins.
-
Bei
der Fluoreszenz-Detektion in vivo in lebenden Zellen eignet sich
insbesondere die Fluoreszenzmikroskopie zur Detektion der Fluoreszenz-Signals.
Weiterhin eignen sich zur Detektion des Fluoreszenz-Signals auch
die Fluoreszenz-Spektroskopie, sowie die Detektion fluoreszierender
Zellen mit Hilfe von FACS.
-
Bei
dem erfindungsgemäßen Verfahren
können
sowohl endogene Proteasen, als auch exogene Proteasen, die in der
Wirtszelle mit Hilfe eines rekombinanten Expressionsvektors exprimiert
werden, detektiert und charakterisiert werden. Der rekombinante
Expressionsvektor umfasst hierbei eine Nukleinsäuresequenz, die die betreffende
Protease unter der Kontrolle eines Promoters kodiert. Unter einer „endogenen
Protease" wird im
folgenden eine Protease verstanden, die in der betreffenden Wirtszelle
selbst entweder konstitutiv, d.h. fortdauernd, oder nicht konstitutiv,
d.h. nur zu bestimmten Zeitpunkten oder unter bestimmten Bedingungen exprimiert
wird. Unter einer „exogenen
Protease" wird im
folgenden eine Protease verstanden, die nicht vom Wirtsgenom selbst
exprimiert wird, sondern mit Hilfe eines von außen durch Transformation eingebrachten
Expressionsvektors in der betreffenden Wirtszelle exprimiert wird.
-
Das
erfindungsgemäße Verfahren
kann allgemein zur Detektion und zur Charakterisierung von Protease-Aktivitäten oder
von Protease-abhängigen
zellulären
Ereignissen eingesetzt werden. Unter einem Protease-abhängigen zellulären Ereignis
sind vorzugsweise auch „natürlich auftretende
Protease-abhängige
Signalkaskaden",
wie insbesondere die Apoptose, die Blutgerinnung, bestimmte entwicklungsspezifische
Signalkaskaden, der Protein-Abbau in der Zelle zu verstehen. Unter
einem Protease-abhängigen
zellulären
Ereignis sind vorzugsweise auch „artifizielle Protease-abhängige Ereignisse" zu verstehen, die
insbesondere dann stattfinden, wenn das Auftreten einer Protease-Aktivität experimentell
von dem Auftreten eines anderen zellulären Ereignisses, wie beispielsweise
einer spezifischen Protein-Protein-Interaktion abhängt.
-
Eine
besonders bevorzugte Abwandlung des erfindungsgemäßen Verfahrens
kann zur Detektion von Protein-Protein-Interaktionen eingesetzt
werden und umfasst die folgenden Verfahrensschritte:
- A) Expression eines ersten und eines zweiten Fusionsproteins
in einer geeigneten Wirtszelle, wobei das erste Fusionsprotein eine
erfindungsgemäße Nukleinsäure kodierend
für ein
nicht fluoreszierendes, aber proteolytisch zur Fluoreszenz aktivierbares
Reporterprotein als erste Domäne
und ein erstes interagierendes Protein als zweite Domäne umfasst,
und wobei das zweite Fusionsprotein eine den flankierenden Protease-Schnittstellen
entsprechende Protease als erste Domäne und ein zweites interagierendes
Protein als zweite Domäne
umfasst,
- B) Interaktion des ersten interagierenden Proteins des ersten
Fusionsprotein mit dem zweiten interagierenden Protein des zweiten
Fusionsprotein,
- C) proteolytische Entfernung der Stör-Aminosäuresequenz aus dem Reporterprotein
des ersten Fusionsproteins durch die Protease des zweiten Fusionsproteins,
- D) Detektion des fluoreszierenden Reporterproteins in Abhängigkeit
von der spezifischen Interaktion zwischen den beiden interagierenden
Domänen
der beiden Fusionsproteine.
-
Die
erfindungsgemäßen Verfahren
eignen sich nicht nur zur reinen Detektion von Protease-Aktivitäten, sondern
auch zur näheren
Charakterisierung von Protease-Aktivitäten oder von Protease-abhängigen Ereignissen.
So können
mit Hilfe der erfindungsgemäßen Verfahren
insbesondere auch solche Substanzen identifiziert werden, die eine
definierte physiologische Protease-Aktivität in der Zelle inhibieren oder
aktivieren. Solche Inhibitoren oder Aktivatoren definierter, physiologisch
relevanter, insbesondere Krankheits-relevanter Protease-abhängiger Ereignisse
könnten
eine signifikante Bedeutung bei der Entwicklung neuer Arzneimittel
haben.
-
Gegenstand
der Erfindung ist daher auch ein Screening-Verfahren zur Identifikation
oder zur Charakterisierung von Inhibitoren oder von Aktivatoren
Protease-abhängiger
Ereignisse in der Zelle. Ein solches Screening-Verfahren könnte die
Verfahrensschritte a) bis d) umfassen, die in einem ersten Versuchsansatz
in Anwesenheit und in einem zweiten Versuchsansatz in Abwesenheit
mindestens einer Test-Substanz, die potentiell Inhibitor- oder Aktivator-Eigenschaften
bezüglich
des getesteten Protease-abhängigen
Ereignisses haben könnte,
durchgeführt
werden. Letztlich kann die Fluoreszenz als Maß der in der Zelle aufgetretenen
Protease-Aktivität
zwischen dem ersten und dem zweiten Versuchsansatz quantitativ verglichen
werden, um die tatsächlichen
Inhibitor- oder Aktivator-Eigenschaften der mindestens einen Test-Substanz
zu beurteilen.
-
Gegenstand
der Erfindung ist auch ein Screening-Verfahren zur Identifikation
oder zur Charakterisierung von Inhibitoren oder von Aktivatoren
definierter Protein-Protein-Interaktionen
in der Zelle. Ein solches Screening-Verfahren könnte die Verfahrensschritte
A) bis D) umfassen, die in einem ersten Versuchsansatz in Anwesenheit
und in einem zweiten Versuchsansatz in Abwesenheit einer oder mehrerer
Test-Substanzen, die potentiell Inhibitor- oder Aktivator-Eigenschaften
bezüglich
einer bestimmten Protein-Protein-Interaktion
haben könnte,
durchgeführt
werden. Letztlich kann die Fluoreszenz als Intensitätsmaß der in
der Zelle aufgetretenen Protein-Protein-Interaktionen zwischen dem
ersten und dem zweiten Versuchsansatz quantitativ verglichen werden,
um die tatsächlichen
Inhibitor- oder Aktivator-Eigenschaften der Test-Substanzen) zu
beurteilen.
-
Ein
weiterer Gegenstand der Erfindung ist ein transgenes nicht menschliches
Tier, welches eine der oben genannten, erfindungsgemäßen Expressionskassetten
in seinem Genom permanent integriert hat. Dieses transgene, nicht
menschliche Tier ist vorzugsweise eine Maus oder eine Ratte. Ein
solches transgenes, nicht menschliches Tier kann vorzugsweise zur
Untersuchung von proteolytischen Vorgängen in vivo, insbesondere
zur Analyse von Caspaseaktivitäten
als Indikator für
apoptotische Vorgänge
eingesetzt werden.
-
Die
Erfindung wird durch das Sequenzprotokoll erläutert, welches die folgenden
Sequenzen umfasst:
SEQ ID NO. 1 enthält ein Fragment des Enhanced
Yellow Fluorescent Protein (EYFP), welches die ersten 15 Aminosäuren des
Proteins aus Aequorea victoria umfasst,
SEQ ID NO. 2 enthält ein Fragment
des Enhanced Yellow Fluorescent Protein (EYFP), welches die ersten
9 Aminosäuren
des Proteins aus Aequorea victoria umfasst,
SEQ ID NO. 3 enthält ein Beispiel
für eine
funktionale Stör-Aminosäuresequenz,
die dazu neigt, α-Helix-Strukturen
auszubilden und gleichzeitig überwiegend
hydrophile Aminosäuren
umfasst (wie insbesondere Glutamat, Aspartat, Arginin und Lysin),
SEQ
ID NO. 4 enthält
ein Beispiel für
eine von Klonierungsstellen und TEV-Protease-Schnittstellen beidseitig flankierte,
funktionale Stör-Aminosäuresequenz,
SEQ
ID NO. 5 enthält
die vollständige
Nukleotidsequenz kodierend für
das Enhanced Yellow Fluorescent Protein (EYFP),
SEQ ID NO.
6 enthält
die vollständige
Aminosäuresequenz
des Enhanced Yellow Fluorescent Proteins (EYFP),
SEQ ID NO.
7 enthält
die vollständige
Nukleotidsequenz des SwitchEYFP-Klons swYFP-CS (Insertion zwischen
die Aminosäuren
6 und 7 von EYFP),
SEQ ID NO. 8 enthält die vollständige Aminosäuresequenz
des SwitchEYFP-Klons swYFP-CS (Insertion zwischen die Aminosäuren 6 und
7 von EYFP),
SEQ ID NO. 9 enthält die vollständige Nukleotidsequenz
des SwitchEYFP-Klons swYFP-H3 (Insertion zwischen die Aminosäuren 9 und
10 von EYFP),
SEQ ID NO. 10 enthält die vollständige Aminosäuresequenz
des SwitchEYFP-Klons swYFP-H3 (Insertion zwischen die Aminosäuren 9 und
10 von EYFP),
SEQ ID NO. 11 enthält den Sinn-Strang des Adapters
aus Beispiel 1,
SEQ ID NO. 12 enthält den Gegensinn-Strang des
Adapters aus Beispiel 1.
-
Die
Erfindung wird weiterhin durch die folgende Zeichnung näher erläutert.
-
1 beschreibt die Klonierungsstrategie
zur Herstellung der Insertionsmutagenese-Bibliothek. In Schritt 1 wird ein Nukleinsäurefragment
kodierend für
EYFP in den Vektor pCMV-EM7-Zeo-βGal
einkloniert. Die Selektion erfolgt hierbei durch die EYFP-Fluoreszenz,
die Resistenz gegen Zeocin und Ampicillin (Amp.). In einem zweiten
Schritt erfolgt die zufällige
Insertion des von PmeI-Schnittstellen flankierten Transposons (TN1-CmR-TN2,
wobei CmR für
das Chloramphenicol-Resistenz-vermittelnde
Gen steht). Die Selektion erfolgt hierbei durch die Resistenz der
Transposon-haltigen Vektoren gegen Chloramphenicol (Cm) und Ampicillin (Amp.).
In Schritt 3 werden die durch Zufallsmutagenese modifizierten Vektoren
mit der Restriktionsendonuklease PmeI geschnitten, wodurch das Transposon
entfernt wird. In den so an zufälliger
Stelle linearisierten Vektor wird ein Adapter umfassend die beidseitig
von TEV-Protease-Schnittstellen flankierte Stör-Aminosäuresequenz einkloniert. Selektioniert
werden Zellen, mit solchen Vektoren, die keine EYFP-Fluoreszenz
mehr zeigen und die eine Resistenz gegen Zeocin aufweisen. In Schritt
4 werden die so selektionierten Vektoren gemeinsam mit Expressionsvektoren,
die die TEV-Protease exprimieren, in PC12-Zellen kotransfiziert.
Es werden hierbei solche Zellen selektioniert, die aufgrund der
proteolytischen Entfernung der Stör-Aminosäuresequenz aus dem EYFP wieder
fluoreszieren.
-
2 zeigt die erste Selektionsphase
der Insertionsmutagenese. In dieser ersten Selektionsphase werden
Expressionsvektoren, die EYFP unter der Kontrolle eines Promoters
exprimieren, hergestellt, wobei die Nukleotidsequenz von EYFP an
einer zufälligen
Stelle von einer Nukleotidsequenz kodierend für eine von TEV-Protease- Schnittstellen beidseitig
flankierten Stör-Aminosäuresequenz
unterbrochen ist (T steht hier bei für eine TEV-Protease-Schnittstelle
und S steht für
die Stör-Aminosäuresequenz).
Hierbei entstehen in Abhängigkeit
von der Position der Insertionsstelle im EYFP entweder fluoreszierende
EYFP-Reporterproteine (in 2 hell
dargestellt) oder nicht fluoreszierende EYFP-Reporterproteine (in 2 grau dargestellt).
-
3 zeigt die zweite Selektionsphase
der Insertionsmutagenese. In dieser zweiten Selektionsphase werden
die in der ersten Selektionsphase aufgrund ihrer Insertionsposition
als nicht fluoreszierend selektionierten Expressionsvektoren gemeinsam
mit einem Expressionsvektor, der die TEV-Protease exprimiert, in PC12-Zellen
kotransfiziert. Durch die hierbei bereitgestellte TEV-Protease-Aktivität in der
Zelle wird die Stör-Aminosäuresequenz
aus dem EYFP-Protein proteolytisch entfernt. Die verbleibenden Domänen des EYFP-Proteins
können
sich hierbei so neu falten bzw. anordnen, daß wieder ein funktionales,
d.h. fluoreszierendes EYFP-Protein entsteht (hell dargestellt in 3) oder auch nicht (dunkel
dargestellt in 3).
-
4 zeigt verschiedene Zellinien,
die mit dem Expressionsvektor des switchEYFP-Klons swYFP-CS transfiziert worden waren
(Spalten, die mit „–TEV" bezeichnet sind)
bzw. mit dem Expressionsvektor des switchEYFP-Klons swYFP-CS und
einem Expressionsvektor, der die TEV-Protease exprimiert, kotransfiziert
worden waren (Spalten, die mit „+TEV" bezeichnet sind). Die 4A bis D zeigen hierbei HEK-Zellen, wobei 4A und 4B Phasenkontrastaufnahmen und 4C und 4D Fluoreszenzaufnahmen derselben Zellen
sind. Die 4E bis 4H zeigen hierbei CHO-Zellen,
wobei 4E und 4F Phasenkontrastaufnahmen
und 4G und 4H Fluoreszenzaufnahmen derselben
Zellen sind. Die 4I und 4J zeigen hierbei COS-Zellen,
wobei 4I eine Phasenkontrastaufnahme
und 4J eine Fluoreszenzaufnahme
derselben Zellen ist. In allen Zellinien zeigt sich, daß die mit dem
Vektor aus dem Klon swYFP-CS transfizierten Zellen in Abwesenheit
der TEV-Protease nicht fluoreszieren, aber in Anwesenheit der TEV-Protease
die Fluoreszenz wiederhergestellt wird. Damit kodiert der Vektor aus
dem Klon swYFP-CS ein erfindungsgemäßes switchEYFP.
-
5 zeigt, daß die erfindungsgemäßen switchEYFPs
auch an definierte Zellkompartimente gekoppelt werden können. Die
obere Reihe von Abbildungen zeigen Phasenkontrastaufnahmen von PC12-Zellen, die
mit einem switchEYFP-Expressionsvektor
und mit einem Expressionsvektor, der die TEV-Protease exprimiert,
kotransfiziert sind. Die untere Reihe von Abbildungen zeigen die entsprechenden
Fluoreszenzaufnahmen. Der SwitchEYFP-Expressionsvektor, mit dem
die Zellen aus Spalte A transfiziert worden waren, ist hierbei der
Expressionsvektor aus dem Klon swYFP-CS. Wie aus dem Vergleich der
Phasenkontrast- und der Fluoreszenzaufnahme aus A ersichtlich ist,
fluoresziert in Spalte A – wie
es zu erwarten wäre – die gesamte
Zelle.
-
Der
SwitchEYFP-Expressionsvektor, mit dem die Zellen aus Spalte B transfiziert
worden waren, entspricht hierbei dem Expressionsvektor aus dem Klon
swYFP-CS mit der Ausnahme, daß das
SwitchEYFP-Protein eine Kernlokalisationsdomäne (nuc) umfasst. Wie aus dem
Vergleich der Phasenkontrast- und der Fluoreszenzaufnahme aus B
ersichtlich ist, fluoresziert in Spalte B – wie es zu erwarten wäre – daher
auch nur der Nucleus der Zelle.
-
Der
SwitchEYFP-Expressionsvektor, mit dem die Zellen aus Spalte C transfiziert
worden waren, entspricht dem Expressionsvektor aus dem Klon swYFP-CS
mit der Ausnahme, daß das
SwitchEYFP-Protein eine Membranlokalisationsdomäne aus dem Membranprotein Syntaxin
(Syx) umfasst. Wie aus dem Vergleich der Phasenkontrast- und der
Fluoreszenzaufnahme aus C ersichtlich ist, fluoresziert in Spalte
C – wie
es zu erwarten wäre – daher
auch nur die Zellmembran der Zelle.
-
6 zeigt, daß nicht
fluoreszierende, proteolytisch zur Fluoreszenz aktivierbare EYFPs
(d.h. switchEYFPs) auch in prokaryontischen Zellen funktionell sind.
Hierzu wurden E.coli-Zellen des Typs DH5α pBAD_TEV mit Expressionsvektoren
kodierend für
switchEYFPs unter der Kontrolle eines Minimalpromoters und mit einem
Vektor, der die TEV-Protease unter der Kontrolle eines mit Arabinose
induzierbaren Promotors exprimiert, kotransfiziert. Anschließend wurde
durch die Zugabe von Arabinose zu den Zellen die Expression der
TEV-Protease induziert. 6 zeigt,
daß nur
solche Zellen, die auf Arabinose-haltigem Medium gewachsen waren
(6A und 6B, „+A" steht für Arabinose-haltiges Medium),
deutlich fluoreszieren, und daß Zellen, die
auf Medium ohne Arabinose gewachsen waren, nicht fluoreszieren (6C und 6D, „–A" steht für Medium ohne Arabinose). Dies
bedeutet, daß auch
in den prokaryontischen E.coli-Zellen die erfindungsgemäßen, nicht fluoreszierenden
switchEYFPs durch Proteolyse zur Fluoreszenz „anschaltbar" sind.
-
7 zeigt die Deletionsmutanten
A bis D des Expressionsvektors aus dem Klon swYFP-CS (Insertionsstelle
zwischen Aminosäure
6 und 7 von EYFP), sowie Fluoreszenzaufnahmen von Zellen, die mit
diesen „deletierten
Expressionsvektoren" transiziert
worden waren (in Abwesenheit der TEV-Protease).
-
Der
Expressionsvektor E entspricht dabei dem Expressionsvektor aus dem
Klon swYFP-CS (Insertionsstelle zwischen Aminosäure 6 und 7 von EYFP).
-
Der
Expressionsvektor D umfasst die Nukleotidsequenz für ein EYFP-Protein
unter der Kontrolle eines Promoters, wobei dieses EYFP-Protein erst
mit der 7. Aminosäure
(E an Position 7) beginnt und zusätzlich an seinem N-Terminus
die folgende Aminosäuresequenz
umfasst: die 11 Aminosäuren „RLMMALLTIHL" der Stör-Aminosäuresequenz
nach SEQ ID No. 3, eine TEV-Protease-Schnittstelle (Aminosäuren „ENLYFQ'G") und die aus der Klonierung resultierenden
Aminosäuren „SGKHE".
-
Der
Expressionsvektor C umfasst die Nukleotidsequenz für ein EYFP-Protein
unter der Kontrolle eines Promoters, wobei dieses EYFP-Protein erst
mit der 7. Aminosäure
(E an Position 7) beginnt und zusätzlich an seinem N-Terminus
die folgende Aminosäuresequenz
umfasst: eine TEV-Protease-Schnittstelle (Aminosäuren „ENLYFQ'G")
und die aus der Klonierung resultierenden Aminosäuren „SGKHE".
-
Der
Expressionsvektor B umfasst die Nukleotidsequenz für ein EYFP-Protein
unter der Kontrolle eines Promoters, wobei dieses EYFP-Protein erst
mit der 7. Aminosäure
(E an Position 7) beginnt und zusätzlich an seinem N-Terminus
mit einer Aminosäuresequenz
umfassend lediglich die aus der Klonierung resultierenden Aminosäuren „SGKHE" fusioniert wird.
-
Der
Expressionsvektor A umfasst die Nukleotidsequenz für ein EYFP-Protein
unter der Kontrolle eines Promoters, wobei dieses EYFP-Protein erst
mit der 7. Aminosäure
(E an Position 7) beginnt und keine zusätzlichen Aminosäuresequenzen
an seinem N-Terminus angehängt
hat.
-
Lediglich
PC12-Zellen, die mit dem Expressionsvektor E oder D transfiziert
worden waren, zeigten keinerlei Fluoreszenz (7D und 7E).
PC12-Zellen, die mit den Expressionsvektoren A, B oder C transfiziert worden
waren, zeigten jedoch immer noch Fluoreszenz (7A, 7B, 7C). Daraus läßt sich
schließen,
daß für den Verlust
der Fluoreszenz der switchEYFPs die Anwesenheit einer Stör-Aminosäuresequenz
notwendig ist, deren minimale Länge
eine Länge
von 10 Aminosäuren
deutlich unterschreitet.
-
8 zeigt die Ergebnisse des
in Beispiel 7 beschriebenen Transkomplementations-Experimentes. Auf
der Ordinate ist die Gesamtfluoreszenz F der positiven Zellen aufgetragen.
Auf der Abzisse steht
„K" für Kontroll-Zellen,
die lediglich mit dem Leervektor transfiziert worden sind,
„A" für Zellen,
die mit einem Expressionsvektor, der eine vollständige TEV-Protease exprimiert, transfiziert worden
sind,
„B" für solche
Zellen, die mit einem ersten und einem zweiten Expressionsvektor
transfiziert worden sind, wobei der erste Expressiosvektor ein Fragment
der TEV-Protease,
umfassend die Aminosäuren
1 bis 118, fusioniert mit der Interaktionsdomäne GCN4 exprimiert, und wobei
der zweite Expressionsvektor ein Fragment der TEV-Protease, umfassend
die Aminosäuren
119 bis 243, fusioniert mit einer weiteren Interaktionsdomäne GCN4
exprimiert,
„C" für solche
Zellen, die mit einem ersten und einem zweiten Expressionsvektor
transfiziert worden sind, wobei der erste Expressionsvektor ein
Fragment der TEV-Protease
umfassend die Aminosäuren
1 bis 118 exprimiert, und wobei der zweite Expressionsvektor ein
Fragment der TEV-Protease umfassend die Aminosäuren 119 bis 243 exprimiert.
-
Alle
Zellen K, A, B, C sind außer
mit den oben bezeichneten Protease-Vektoren auch mit einem Expressionsvektor,
der ein erfindungsgemäßes switchEYFP
exprimiert, kotransfiziert.
-
8 zeigt, daß die Aktivität einer
Protease – und
die dadurch bedingte Wiederherstellung der Fluoreszenz nach proteolytischer
Entfernung der von TEV-Protease-Schnittstellen
flankierten Stör-Aminosäuresequenz – sowohl
durch eine intakte, vollständige
TEV-Protease (siehe A aus 8)
als auch durch eine Protein-Protein-Interaktions-abhängige Transkomplementation
einer funktionalen TEV-Protease
bereitgestellt werden kann (siehe B aus 8). Damit läßt sich das erfindungsgemäße Verfahren
auch zur Detektion und Charakterisierung von Protein-Protein-Interaktionen,
sowie zum Screening von Inhibitoren oder Aktivatoren einer Protein-Protein-Interaktion
verwenden.
-
Die
Erfindung wird durch die folgenden experimentellen Beispiele näher erläutert.
-
Beispiel 1: Herstellung
bzw. Screening nach nicht fluoreszierenden, aber proteolytisch zur
Fluoreszenz aktivierbaren EYFP-Varianten durch Insertionsmutagenese
-
Zur
Herstellung nicht fluoreszierender, aber proteolytisch zur Fluoreszenz
aktivierbarer EYFP-Reporterproteine wurde das offene Leseraster
des Enhanced Yellow Fluorescent Protein (EYFP) mit den Oligonukleotid-Primern
EYFP-Xhol-sense (Seq: gggctcgagaccatggtgagcaagggcgagga) und EYFP
Xhol-antisense (Seq: gggctcgagcttgtacagctcgtccatgccga) mittels PCR
amplifiziert und in den rekombinanten Vektor pCMV-EM7-Zeo-βGal kloniert.
Der aus dieser Klonierung hervorgegangene rekombinante Vektor wird
im folgenden als pCMV-EYZG bezeichnet. Der Vektor enthält den CMY-Promoter,
eine SV40-Polyadenylierungssequenz sowie den bakteriellen Minimalpromoter
EM7 zur Expression nachgeschalteter Gene in E.coli und in höheren eukaryontischen
Zellen. Das Transkript des pCMV-EYZG kodiert für ein Fusionsprotein aus drei
Domänen,
nämlich
aus EYFP, aus dem ZeoR-Protein, einem Protein, das Resistenz gegen
Zeocin vermittelt, und aus dem β-Galaktosidase-Protein
von E.coli. Die zur Klonierung verwendeten Schnittstellen waren
hierbei so gewählt,
dass ein durchgehendes Leseraster für alle drei Domänen des
Fusionsproteins generiert wurde. (siehe 1). Die Funktionalität aller drei Einzelkomponenten
des Fusionsproteins wurde durch Analyse der Epifluoreszenz in Bakterien
und in eukaryontischen Zellen, durch Selektion mit Zeocin und durch
einen kolorimetrischen β-Gal-Nachweis
erbracht. Weiterhin wurde in den Vektor pCMV-EM7-Zeo-βGal ein EYFP-Leseraster
in gegenläufiger
Orientierung in Bezug auf das ZeoR-βGal-Leseraster als Kontrollvektor
kloniert. Nach Transformation dieses Kontrollvektors in E.coli-Zellen
oder auch nach Transfektion dieses Kontrollvektors in eukaryontische
Zellen konnte weder Epifluoreszenz nachgewiesen, noch eine Resistenz
gegen Zeocin vermittelt werden, was mit hoher Wahrscheinlichkeit
durch die Unterbrechung des Leserasters des EYZG-Fusionsproteins
begründet
ist. Trotz der Unterbrechung des Leserasters des EYZG-Fusionsproteins
konnte jedoch eine deutliche Aktivität der β-Galaktosidase im kolorimetrischen
Test nachgewiesen werden. Daher wurden rekombinante Klone im nachfolgend
beschriebenen Klonierungsverfahren zunächst mit Zeocin selektioniert
und nachfolgend deren Epifluoreszenz analysiert.
-
Die
Insertionsmutagenese zur Herstellung nicht fluoreszierenden, aber
proteolytisch zur Fluoreszenz aktivierbaren EYFP-Varianten wurde über die
folgenden Verfahrensschritte durchgeführt:
- 1)
Mit dem Plasmid pCMV-EYZG wurde eine Transposon-vermittelte Zufallsmutagenese
mit Hilfe des GPS-LS Kit von New England Biolabs (NEB) nach Angaben
des Herstellers durchgeführt.
Hierbei wurden zunächst
E.coli-Zellen mit dem Target-Plasmid pCMV-EYZG und mit dem Transprimer-Donor-Plasmid
aus dem Kit transformiert. Durch eigentliche Mutagenese erfolgte
durch die Anwesenheit der TnsABC-Transposase
in der Zelle. Aus der Mutagenese gingen etwa 500 Ampicillin-, Zeocin-
und Chlormphenicol-resistente rekombinante Bakterienklone hervor
und wurden nach Kultivierung auf entsprechenden LB-Selektionsplatten
amplifiziert. Etwa 1000 Bakterienkolonien der amplifizierten rekombinanten
Bakterienklone wurden vereinigt und die Plasmid-DNA wurde nach Standardverfahren
(Qiaprep, Qiagen) isoliert.
Die Selektion auf Ampicillin-Resistenz
garantiert hierbei, daß das
Target-Plasmid in der Zelle anwesend ist. Die Selektion auf Zeocin-Resistenz
garantiert hierbei, daß die
Insertion des Transposons nicht zu einer Verschiebung des Leserasters
geführt
hat. Die Selektion auf Chloramphenicol-Resistenz garantiert hierbei, daß ein Transposon
im Target-Plasmid anwesend ist.
- 2) Die in Schritt 1 gewonnene komplexe Plasmid-DNA-Mischung
wurde mit dem Restriktionsenzym XhoI (NEB) verdaut. Nach gelelektrischer
Auftrennung wurde ein 1,9 kb langes Fragment umfassend das EYFP mitsamt
zufällig
inseriertem Transposon aus dem Gel eluiert (Qiaprep, Qiagen) und
anschließend
mit einem XhoI-linearisierten, dephosphorylierten pCMV-EM7 ZeoR-βGal-Vektor
ligiert. Die Ligationsprodukte wurden in E. coli-Zellen transformiert
und auf Ampicillin- und Chloramphenicol-Resistenz selektioniert.
Etwa 5000 erfolgreich selektionierte Bakterienkolonien wurden vereinigt.
Anschließend
wurde aus diesen Kolonien Plasmid-DNA nach Standardverfahren (Qiaprep,
Qiagen) isoliert.
- 3) Die in Schritt 2 gewonnene komplexe Plasmid-DNA-Mischung
wurde mit dem Restriktionsenzym PmeI (NEB) verdaut. Durch diesen
Restriktionsschnitt wird ein großes Fragment aus dem Transposon
entfernt, so daß nach
dem PmeI-Restriktionsschnitt nur noch wenige flankierende Basen
des Transposons, nämlich 15
bp (5 Aminosäuren
auf Protein-Ebene, siehe Produktbeschreibung des Herstellers, GPS-LS
Kit, NEB) an der Insertionsstelle verbleiben. Die Produkte wurden
anschließend
mit alkalischer Phosphatase behandelt und gelelektrophoretisch aufgetrennt.
Die an zufälligen
Stellen des EYFP-Leserasters
lineariserten Vektoren wurden aus dem Gel nach Standardmethoden
isoliert (Qiaquick, Qiagen).
- 4) Es wurde ein Adapter mit einer von TEV-Protease-Schnittstellen
flankierten Stör-Nukleotidsequenz
kodierend für
eine Stör-Aminosäuresequenz
durch Hybridisierung der partiell überlappenden 5' phosphorylierten
Oligonukleotide
mit der Sequenz 5'P-agcggtgaaaacctatacttccaaggcctgctggcactgctgatgatg
aaggccctgctgcgccgcctgctgaaggcact-3' (SEQ ID NO. 11) und
mit der Sequenz
5'-accgctaccttggaagtacaggttttctaaatgaatagtcagcagtgccatcatcagc-3' (SEQ ID NO. 12)
und durch Auffüllen
der einzelsträngigen
Bereiche mit dem Klenow-Enzym (Roche) hergestellt.
Die flankierenden „TEV-Protease-Schnittstellen" stellen hierbei
spezifische Erkennungs- und
Spaltstellen der NIa TEV-Protease dar.
Das Stör-Fragment
kodiert in der angegebenen Orientierung für eine von TEV-Schnittstellen flankierte,
starre, partiell geladene α-Helix.
Die Analyse der vermutlichen Sekundärstruktur des Stör-Fragments
wurde mit dem Programm Predator (EMBL) durchgeführt.
Die linearisierten,
dephosphorylierten Vektoren aus Schritt 3 wurden mit dem 5'-phosphorylierten Adapter mit der von
TEV-Protease-Schnittstellen flankierten Stör-Nukleotidsequenz, im folgenden als 'Stör-Fragment' bezeichnet, ligiert
(siehe Beispiel 1, Nr. 4).
Die Ligationsprodukte wurden in
E.coli-Zellen transformiert. Rekombinante Klone wurden mit Ampicillin
und Zeocin selektioniert. 480 erfolgreich selektionierte Bakterienkolonien
wurden einzeln amplifiziert. Anschließend wurde die Plasmid-DNA
der Einzelklone nach Standardverfahren (Qiaprep, Qiagen) isoliert.
- 5) Die Plasmid-DNA der Einzelklone aus Schritt 4 wurde anschließend in
PC12-Zellen analysiert.
Für jeden Einzelklon
wurden jeweils zwei Transformationsansätze durchgeführt. In
dem ersten Transformationsansatz wurden je 1 μl der Plasmid-Präparationen
mit Lipofectamin 2000 (Invitrogen) in PC12-Zellen transfiziert.
In einem zweiten Transformationsansatz wurden je 1 μl der Plasmid-Präparationen
und 0,1 μg
eines Expressionsvektors, der die TEV-Protease unter der Kontrolle
eines Promoters exprimieren kann (pCMV-TEV), mit Lipofectamin 2000
(Invitrogen) in PC12-Zellen transfiziert. Anschließend wurde überprüft, ob die
transformierten PC12-Zellen des jeweils ersten und des jeweils zweiten
Transformationsansatzes fluoreszieren oder nicht. Selektioniert
wurden solche Klone, die in Abwesenheit der TEV-Protease (erster Transformationsansatz)
aufgrund ihrer Insertion der Stör-Aminosäuresequenz
im EYFP nicht fluoreszieren, aber in Gegenwart der TEV-Protease
(zweiter Transformationsansatz) aufgrund der proteolytischen Abspaltung
der Stör-Aminosäuresequenz
aus dem EYFP fluoreszieren. Bei zwei Klonen konnte tatsächlich ohne
TEV-Protease-Aktivität
keine Epifluoreszenz detektiert werden, nach Kotransfektion der
entsprechenden Plasmid-DNA und des TEV-Protease exprimierenden Vektors konnten
jedoch fluoreszierende Zellen detektiert werden. Diese beiden Klone
wurden als swYFP-H3 und swYFP-CS bezeichnet.
-
Das
aus dem Klon swYFP-CS hervorgehende EYFP-Protein besitzt eine von
TEV-Protease-Schnittstellen
flankierte Stör-Aminosäuresequenz,
die zwischen den Aminosäuren
6 und 7 inseriert worden ist. Das aus dem Klon swYFP-H3 hervorgehende
EYFP-Protein besitzt eine von TEV-Protease-Schnittstellen flankierte Stör-Aminosäuresequenz,
die zwischen den Aminosäuren
9 und 10 inseriert worden ist.
-
Somit
scheint der N-Terminus des EYFP für die Insertion bzw. für das Anhängen einer
Stör-Aminosäuresequenz
zur Generierung eines nicht fluoreszierenden, aber proteolytisch
zur Fluoreszenz aktivierbaren Reporterproteins geeignet zu sein.
Eine solche Stör-Aminosäuresequenz-Insertion,
die als proteolytischer Schalter zwischen Fluoreszenz oder Nicht-Fluoreszenz
eines Reporterproteins fungiert, kann für zahlreiche Assay-Varianten
zur Detektion und/oder zur Charakterisierung von Protease-Aktivitäten oder
von Proteaseabhängigen
Ereignissen eingesetzt werden.
-
Beispiel 2: Feinanalyse
der Insertionsstelle und der Länge
der Killersequenz
-
Der
kritische Bereich für
die Insertion der Schaltersequenz ist der N-Terminus von EYFP. Zur
genauen Bestimmung, welche Insertions-Positionen sich besonders
zum „Ab-
und Anschalten" der
Fluoreszenz des Reporterproteins eignen, wurde die von TEV-Protease-Schnittstellen flankierte
Stör-Aminosäuresequenz
in getrennten Experimenten nach jeder der ersten 12 Aminosäuren des
Proteins EYFP insertiert. Die so hergestellten EYFP-Varianten wurden
anschließend
durch Kotransfektion mit einem Vektor, der die TEV-Protease exprimiert,
in PC12-Zellen getestet.
-
Tabelle
2 zeigt das Ergebnis dieser Experimente: so steht beispielsweise „switchEYFP_1_Störseq._2" für einen
Expressionsvektor, der ein switchEYFP unter der Kontrolle eines
Promoters umfasst, wobei das switchEYFP eine Insertion einer beidseitig
von TEV-Protease-Schnittstellen flankierte Stör-Aminosäuresequenz nach SEQ ID No.
3 enthält.
Das Ergebnis, welches in Tabelle 2 zusammengefaßt ist, zeigt, daß prinzipiell
bei allen Insertionen zwischen den Aminosäuren 2 und 9 EYFP-Varianten
entstehen, die proteolytisch „anschaltbar" bezüglich ihrer
Fluoreszenz sind, d.h. diese Varianten führen in Abwesenheit einer Protease-Aktivität nicht zur
Fluoreszenz, aber in Gegenwart einer Protease-Aktivität zur Fluoreszenz.
Bei EYFP-Varianten, die zu einer weiteren Verkürzung von EYFP führen (10/11,
11/12 etc.), funktioniert das „proteolytische
Anschalten" der
Fluoreszenz des Proteins weniger gut, da die Minimalsequenz zur
Ausbildung der Fluoreszenz unterschritten wird (Li et al.; J Biol
Chem. 1997 Nov 7;272(45):28545-9).
-
Es
konnte weiterhin gezeigt werden, daß alternativ zur Insertion
an beliebiger Position zwischen den Aminosäuren 2 und 9 die von TEV-Protease-Schnittstellen
flankierte Stör-Aminosäuresequenz
auch einfach an den N-Terminus des EYFPs angehängt werden kann. Hierbei können die
ersten 1 bis 9 Aminosäuren
des EYFPs auch durchaus deletiert und die flankierte Stör-Aminosäuresequenz
direkt an den N-Terminus des entsprechenden restlichen EYFP-Fragments
fusioniert sein.
-
-
Beispiel 3: Unterschiedlich
lokalisierte Varianten von nicht fluoreszierenden, aber proteolytisch
zur Fluoreszenz aktivierbaren Reporterproteinen
-
Nicht
fluoreszierende, aber proteolytisch zur Fluoreszenz aktivierbare
Reporterproteine, die eine ein- oder zweiseitig von Protease-Schnittstellen
flankierte Stör-Aminosäuresequenz
am N-Terminus angehängt oder
an den obigen Positionen insertiert haben, lassen sich auch durch
Fusion an andere Proteine oder Proteindomänen an verschiedenen Stellen
innerhalb der Zelle lokalisieren. So konnten beispielsweise eine switch-EYFP-Variante
mit Kernlokalisation und eine switch-EYFP-Variante mit Membranverankerung
in PC12-Zellen erfolgreich getestet werden.
-
Beispiel 3.1: Nicht fluoreszierendes,
proteolytisch zur Fluoreszenz aktivierbares EYFP mit Kernlokalisation
-
An
den C-Terminus eines switch-EYFPs wurde ein Kernlokalisationssignal
im Leseraster fusioniert (im folgenden „switchEYFPnuc" genannt). Dies führte dazu,
daß das
Protein bevorzugt in den Zellkern von eukaryontischen Zellen transportiert
wird. In 5B werden PC12-Zellen
gezeigt, welche mit einem Expressionsvektor, der switchEYFPnuc exprimiert,
sowie mit einem Expressionsvektor, der die TEV-Protease exprimiert, kotransfiziert
wurden. Es konnte eine von der Anwesenheit der TEV-Protease abhängige Fluoreszenz
im Zellkern beobachtet werden.
-
Beispiel 3.2: Nicht fluoreszierendes,
proteolytisch zur Fluoreszenz aktivierbares EYFP mit Membranlokalisation
-
Ein
switchEYFP wurde an den N-Terminus eines Fragmentes des synaptischen
Membranproteins Syntaxin 1A (Aminosäure 1–147) fusioniert (im folgenden „switchEYFPsyx" genannt). Aufgrund
der C-terminalen Transmembrandomäne
wird das Fusionsprotein an der Zellmembran von eukaryontischen Zellen
verankert. In 5C werden
PC12-Zellen gezeigt, welche mit einem Expressionsvektor, der switchEYFPsyx
exprimiert, sowie mit einem Expressionsvektor, der die TEV-Protease
exprimiert, kotransfiziert wurden. Es konnte eine von der Anwesenheit
der TEV-Protease abhängige
Fluoreszenz hauptsächlich
an der Peripherie der Zelle beobachtet werden. Diese Variante switchEYFPsyx
läßt sich
besonders gut durch Kotransfektion einer ebenfalls membranverankerten
TEV-Protease aktivieren (Daten nicht gezeigt).
-
Beispiel 4: Expression
von nicht fluoreszierenden, proteolytisch zur Fluoreszenz aktivierbaren
EYFP in weiteren eukaryontischen Zellen
-
Die
Funktion der switchEYFPs wurden zusätzlich in weiteren Zelllinien
getestet. In 4 wird
gezeigt, daß die
durch Proteolyse anschaltbare Fluoreszenz des EYFPs auch in CHO- Zellen (Chinese hamster
ovary-Zellen) und in HEK293-Zellen (Human embryonic Kidney-Zellen) beobachtet
werden kann. In allen getesteten Zelllinien waren die getesteten
switchEYFPs nicht-fluoreszent und gewannen ihre Fluoreszenz erst
durch die Kotransfektion eines Expressionsvektors kodierend für die TEV-Protease
zurück.
-
Beispiel 5: Expression
von nicht fluoreszierenden, proteolytisch zur Fluoreszenz aktivierbaren
EYFP in Prokaryonten
-
Es
sollte untersucht werden, ob nicht fluoreszierende, proteolytisch
zur Fluoreszenz aktivierbare EYFPs (d.h. switchEYFPs) auch in prokaryontischen
Zellen funktionell sind. Hierzu wurden E.coli-Zellen des Typs DH5αpBAD_TEV
mit Expressionsvektoren kodierend für switchEYFPs transformiert. DH5αpBAD_TEV-Zellen
enthalten ein Plasmid, welches die TEV-Protease unter der Kontrolle
eines mit Arabinose induzierbaren Promotors exprimiert. Dieses Plasmid
besitzt weiterhin einen „p15A-origin", so das Kotransformation
mit „ColE1-Origin-Vektoren" möglich sind.
-
Diese
E.coli-Zellen wurden mit einem Expressionsvektor kodierend für ein switchEYFP
unter der Kontrolle des EM7-Minimalpromotors transformiert. Anschließend wurde
durch die Zugabe von Arabinose zu den Zellen die Expression der
TEV-Protease induziert. In 6 wird
gezeigt, daß nur
solche Zellen, die auf Arabinose-haltigem Medium gewachsen waren,
deutlich grün
fluoreszieren. Dies bedeutet, daß auch in den prokaryontischen
E. coli-Zellen die erfindungsgemäßen nicht
fluoreszierenden switchEYFPs durch Proteolyse zur Fluoreszenz „anschaltbar" sind.
-
Beispiel 6: Minimale Länge der
von TEV-Protease-Schnittstellen flankierten Stör-Aminosäuresequenz
-
Bei
den im Rahmen des Screening-Verfahrens (siehe Beispiel 1) zunächst identifizierten
switchEYFP-Klonen swYFP-CS (Stör-Aminosäuresequenz
insertiert zwischen Aminosäure
6 und 7 von EYFP) und swYFP-H3 (Stör-Aminosäuresequenz insertiert zwischen
Aminosäure
9 und 10 von EYFP) sollte überprüft werden,
welcher Anteil der Stör-Aminosäuresequenz
für den
Verlust der Fluoreszenz des EYFPs verantwortlich ist, bzw. wie lang
die Stör-Aminosäuresequenz
sein muß,
um ihren Zweck, nämlich
die Zerstörung
der EYFP-Fluoreszenz
und die Wiederherstellung der EYFP-Fluoreszenz nach ihrer proteolytischen Entfernung, zu
erfüllen.
-
Dazu
wurden die Deletionsmutanten A bis D des Vektors aus dem Klon swYFP-CS
hergestellt. Bei diesen Deletionsmutanten wurden jeweils Teile der
Stör-Aminosäuresequenz
nach SEQ ID No. 3, gemeinsam mit einer TEV-Protease-Schnittstelle
und der GPS-Linker Sequenz (SGKHE), an ein EYFP-Fragment angehängt, welches
erst mit der 7. Aminosäure
der EYFP-Sequenz beginnt (siehe 7A bis E).
-
Die
Variante E aus 7 entspricht
hierbei dem Expressionsvektor aus dem Klon swYFP-CS, d.h. in diesem
Expressionsvektor ist die gesamte Stör-Aminosäuresequenz nach SEQ ID No.
3, gemeinsam mit einer TEV-Protease-Schnittstelle und der GPS-Linker
Sequenz nach der Aminosäure
6 der EYFP-Aminosäuresequenz
insertiert. Wie aus 7E ersichtlich,
zeigen Zellen, die mit einem Expressionsvektor der Variante E transformiert
wurden, keinerlei Fluoreszenz.
-
Bei
der Variante D des Expressionsvektors aus 7 wurde lediglich eine Aminosäuresequenz
umfassend die letzten 11 Aminosäuren
der Stör-Aminosäuresequenz
nach SEQ ID No. 3, die TEV-Schnittstelle und die GPS-Linkersequenz
N-terminal an ein EYFP-Fragment angehängt, welches erst mit der 7.
Aminosäure der
EYFP-Sequenz beginnt. 7 D
zeigt, daß Zellen,
die mit einem Expressionsvektor der Variante D transformiert wurden,
ebenfalls nicht fluoreszieren.
-
Bei
der Variante C des Expressionsvektors wurde nur die TEV-Schnittstelle
mit der GPS-Linkersequenz
und bei der Variante B des Expressionsvektors lediglich die GPS-Linkersequenz alleine
N-terminal an ein EYFP-Fragment ankloniert, welches erst mit der
7. Aminosäure
der EYFP-Sequenz beginnt. Weiterhin wurde eine Variante A des Expressionsvektors
hergestellt, die keine zusätzliche
Sequenz trägt,
die jedoch erst bei Aminosäure
E7 startet (7).
-
Die
Konstrukte A, B, C, D, E wurden durch Transfektion in PC12-Zellen
getestet und mikroskopisch analysiert. 7A bis E zeigen
die Fluoreszenz-Aufnahmen der mit den entsprechenden Expressionsvektoren A
bis E transfizierten Zellen 24 Stunden nach der Transfektion. Lediglich
die Zellen, die entweder mit einem Expressionsvektor umfassend EYFP
mit der gesamten Stör-Aminosäuresequenz
(Variante E) transfiziert worden waren oder die mit einem Expressionsvektor
umfassend EYFP mit einem 11 Aminosäuren langen Fragment der Stör-Aminosäuresequenz
(Variante D) transfiziert worden waren, zeigten keinerlei Fluoreszenz.
Zellen, die mit den übrigen
Expressionsvektoren (Varianten A, B, C) transfiziert worden waren
und die demnach keine Stör-Aminosäuresequenz
umfassen, zeigten hingegen auch in Abwesenheit einer Protease-Aktivität Fluoreszenz.
Bei Zellen, die mit der Expressionsvektor D transfiziert worden
waren („Expressionsvektor
umfassend EYFP mit einem 11 Aminosäuren langen Fragment der Stör-Aminosäuresequenz"), war jedoch 24
Stunden nach der Transfektion – im
Vergleich zu Zellen, die mit Expressionsvektor E transfiziert worden
waren – eine
schwache Fluoreszenz der transfizierten Zellen zu beobachten. Daraus
läßt sich
schließen,
daß für den Verlust
der Fluoreszenz der switchEYFPs die Anwesenheit einer Stör-Aminosäuresequenz
notwendig ist, deren minimale Länge
eine Länge
von 10 Aminosäuren
deutlich unterschreitet. Die erfindungsgemäßen Stör-Aminosäuresequenzen sollten daher
eine Länge
von mindestens 3 Aminosäuren,
vorzugsweise von mindestens 5 Aminosäuren, bevorzugter von 7 Aminosäuren, insbesondere
von 10 Aminosäuren
umfassen.
-
Beispiel 7: SwitchEYFP
als Interaktions-Sensor
-
Ein
erfindungsgemäßes SwitchEYFP
kann insbesondere auch als Indikator für Protein-Protein Interaktionen eingesetzt werden.
Dabei sollte die spezifische Interaktion zwischen zwei Proteinen,
zwischen einem Protein und einer Substanz oder zwischen zwei Proteinen
und einer Substanz zur Aktivierung einer proteolytischen Aktivität führen. Diese
Kopplung zwischen einer Protein-Protein-Interaktion und einer proteolytischen Aktivität kann vorzugsweise
durch die Protein-Protein-Interaktions-abhängige Transkomplementation
einer funktionalen Protease erreicht werden, wie in der prioritätsfrüheren und
nachveröffentlichen
Anmeldung (interne Bezeichnung
DE
102 11 063.8 ) beschrieben. Hierbei werden zwei potentiell
miteinander interagierende Proteine an jeweils ein nicht funktionales
Fragment einer Protease, insbesondere der TEV-Protease (243 Aminosäuren lang)
fusioniert unter Generierung zweier Fusionsproteine. Durch die Interaktion
dieser Proteine, werden die Proteasefragmente in große räumliche
Nähe zueinander
gebracht, so daß die
verlorengegangene Protease-Aktivität zurückerlangt wird.
-
Hierbei
kann das erste Fusionsprotein insbesondere ein Fragment umfassend
die Aminosäuren
1 bis 118 der TEV-Protease und ein erstes potentiell interagierendes
Protein umfassen und das zweite Fusionsprotein kann insbesondere
ein Fragment umfassend die Aminosäuren 119 bis 243 der TEV-Protease
und ein zweites potentiell interagierendes Protein umfassen. Eine
Teilung der TEV-Protease zwischen den Aminosäuren 118 und 119 zur Transkomplementation
hat sich experimentell als günstig
erwiesen, d.h. die hierbei generierten TEV-Protease-Fragmente sind
für sich
alleine nicht funktionell, bilden jedoch eine funktionale TEV-Protease,
sofern diese sich durch die Interaktion der interagierenden Domänen der
Fusionproteine räumlich
aneinander annähern.
Es sind jedoch auch Fusionsproteine mit anderen Proteasefragmenten
als den oben beschriebenen oder auch Fusionsproteine mit überlappenden
Proteasefragmenten zur Bereitstellung einer Protease-Aktivität durch
Transkomplementation fähig.
-
Im
Rahmen dieses Beispiels sollte die nach Transkomplementation zweier
Proteasefragmente wiedererlangte Protease-Aktivität mit Hilfe
eines erfindungsgemäßen switchEYFPs
als Detektor nachgewiesen werden. Als Modell wurden die Interaktionsdomänen des
GCN4-Leuzin-Zipper-Proteins verwendet, an deren C-Terminus die Protease-Fragmente
Aminosäure
1 bis 118 und Aminosäure
119 bis 243 fusioniert worden waren. Die Interaktionsdomänen des
GCN4-Leuzin-Zipper-Proteins sind dafür bekannt, Homodimere auszubilden.
Die oben genannten Konstrukte, sowie eine positive und eine negative
Kontrolle wurden jeweils gemeinsam mit einem Expressionsvektor,
der eine beidseitig durch TEV-Protease-Schnittstellen flankierte
Stör-Aminosäuresequenz,
welche nach der Aminosäure
6 von EYFP insertiert ist (switchEYFP_6_Killer_7), in PC12-Zellen
kotransfiziert.
-
8 zeigt das Ergebnis nach
Auswertung der Fluoreszenz mittels FACS-Analyse. Die Kotransfektion der
gesamten („ungeteilten") TEV-Protease und
eines switchEYFPs führt
zu einer Protease-Aktivität
in der Zelle, zur proteolytischen Entfernung der Stör-Aminosäuresequenz
und damit schließlich
zur Fluoreszenz der Zelle. Die Kotransfektion eines switchEYFP und
von Teilfragmenten der TEV-Protease jeweils fusioniert an die Interaktionsdomänen von
GCN4 führt
ebenfalls zu einer Protease-Aktivität in der Zelle, zur proteolytischen
Entfernung der Stör-Aminosäuresequenz
und letztlich zur Fluoreszenz der Zelle. Keine Aktivierung erfolgt
bei der Kotransfektion eines switchEYFP und eines Leervektors bzw.
der Vektors umfassend die TEV-Fragmente alleine ohne die Interaktionsdomänen von
GCN4 (Negativ-Kontrollen).
-
-
-
-
-
-
-
-
-
-
-
-
-
-