RU2736728C2 - Транспозиция с сохранением сцепления генов - Google Patents
Транспозиция с сохранением сцепления генов Download PDFInfo
- Publication number
- RU2736728C2 RU2736728C2 RU2019138705A RU2019138705A RU2736728C2 RU 2736728 C2 RU2736728 C2 RU 2736728C2 RU 2019138705 A RU2019138705 A RU 2019138705A RU 2019138705 A RU2019138705 A RU 2019138705A RU 2736728 C2 RU2736728 C2 RU 2736728C2
- Authority
- RU
- Russia
- Prior art keywords
- sequence
- nucleic acid
- target nucleic
- dna
- barcode
- Prior art date
Links
- 230000017105 transposition Effects 0.000 title claims description 28
- 108090000623 proteins and genes Proteins 0.000 title abstract description 38
- 238000004321 preservation Methods 0.000 title description 3
- 150000007523 nucleic acids Chemical class 0.000 claims abstract description 269
- 102000039446 nucleic acids Human genes 0.000 claims abstract description 209
- 108020004707 nucleic acids Proteins 0.000 claims abstract description 209
- 239000007787 solid Substances 0.000 claims abstract description 113
- 108091034117 Oligonucleotide Proteins 0.000 claims abstract description 83
- 239000000203 mixture Substances 0.000 claims abstract description 57
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 claims abstract description 48
- 230000000295 complement effect Effects 0.000 claims abstract description 39
- 239000000969 carrier Substances 0.000 claims abstract description 21
- 238000012163 sequencing technique Methods 0.000 claims description 79
- 108010020764 Transposases Proteins 0.000 claims description 68
- 102000008579 Transposases Human genes 0.000 claims description 67
- 239000012634 fragment Substances 0.000 claims description 61
- 238000013467 fragmentation Methods 0.000 claims description 14
- 238000006062 fragmentation reaction Methods 0.000 claims description 14
- 108010012306 Tn5 transposase Proteins 0.000 claims description 10
- 239000004005 microsphere Substances 0.000 claims description 7
- 239000002245 particle Substances 0.000 claims description 4
- 230000011987 methylation Effects 0.000 abstract description 18
- 238000007069 methylation reaction Methods 0.000 abstract description 18
- 230000000694 effects Effects 0.000 abstract description 7
- 239000000126 substance Substances 0.000 abstract description 6
- 108020004414 DNA Proteins 0.000 description 184
- 238000000034 method Methods 0.000 description 152
- 238000006243 chemical reaction Methods 0.000 description 86
- LSNNMFCWUKXFEE-UHFFFAOYSA-M Bisulfite Chemical compound OS([O-])=O LSNNMFCWUKXFEE-UHFFFAOYSA-M 0.000 description 55
- 239000011324 bead Substances 0.000 description 40
- 239000000523 sample Substances 0.000 description 30
- HEMHJVSKTPXQMS-UHFFFAOYSA-M Sodium hydroxide Chemical compound [OH-].[Na+] HEMHJVSKTPXQMS-UHFFFAOYSA-M 0.000 description 21
- 210000004027 cell Anatomy 0.000 description 21
- 210000002381 plasma Anatomy 0.000 description 20
- 102100033215 DNA nucleotidylexotransferase Human genes 0.000 description 19
- 238000010586 diagram Methods 0.000 description 19
- 108010090804 Streptavidin Proteins 0.000 description 18
- 238000001514 detection method Methods 0.000 description 18
- 238000012408 PCR amplification Methods 0.000 description 17
- 238000003556 assay Methods 0.000 description 17
- 230000015572 biosynthetic process Effects 0.000 description 17
- 238000009396 hybridization Methods 0.000 description 17
- 239000000047 product Substances 0.000 description 17
- 230000003321 amplification Effects 0.000 description 16
- 238000003199 nucleic acid amplification method Methods 0.000 description 16
- 102000053602 DNA Human genes 0.000 description 15
- 108091028043 Nucleic acid sequence Proteins 0.000 description 13
- 229960002685 biotin Drugs 0.000 description 13
- 239000011616 biotin Substances 0.000 description 13
- 238000012217 deletion Methods 0.000 description 13
- 230000037430 deletion Effects 0.000 description 13
- 239000002773 nucleotide Substances 0.000 description 13
- 125000003729 nucleotide group Chemical group 0.000 description 13
- 238000004458 analytical method Methods 0.000 description 12
- 230000008901 benefit Effects 0.000 description 12
- 230000001413 cellular effect Effects 0.000 description 12
- 239000000243 solution Substances 0.000 description 12
- 210000000349 chromosome Anatomy 0.000 description 11
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N (+)-Biotin Chemical compound N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 description 10
- 210000004369 blood Anatomy 0.000 description 10
- 239000008280 blood Substances 0.000 description 10
- 238000009826 distribution Methods 0.000 description 10
- 201000004283 Shwachman-Diamond syndrome Diseases 0.000 description 9
- 238000002360 preparation method Methods 0.000 description 9
- 108020004635 Complementary DNA Proteins 0.000 description 8
- 108700024394 Exon Proteins 0.000 description 8
- 108091008109 Pseudogenes Proteins 0.000 description 8
- 102000057361 Pseudogenes Human genes 0.000 description 8
- 238000010804 cDNA synthesis Methods 0.000 description 8
- 239000002299 complementary DNA Substances 0.000 description 8
- 108010014303 DNA-directed DNA polymerase Proteins 0.000 description 7
- 102000016928 DNA-directed DNA polymerase Human genes 0.000 description 7
- 108010033040 Histones Proteins 0.000 description 7
- 108010061833 Integrases Proteins 0.000 description 7
- 239000000872 buffer Substances 0.000 description 7
- 229920001519 homopolymer Polymers 0.000 description 7
- 239000000178 monomer Substances 0.000 description 7
- 235000018102 proteins Nutrition 0.000 description 7
- 102000004169 proteins and genes Human genes 0.000 description 7
- 239000007790 solid phase Substances 0.000 description 7
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 6
- 102100034343 Integrase Human genes 0.000 description 6
- 206010028980 Neoplasm Diseases 0.000 description 6
- 238000003780 insertion Methods 0.000 description 6
- 230000037431 insertion Effects 0.000 description 6
- 238000002372 labelling Methods 0.000 description 6
- 238000004519 manufacturing process Methods 0.000 description 6
- 238000002156 mixing Methods 0.000 description 6
- 239000012071 phase Substances 0.000 description 6
- 108010008286 DNA nucleotidylexotransferase Proteins 0.000 description 5
- 101150116023 TNP1 gene Proteins 0.000 description 5
- 235000020958 biotin Nutrition 0.000 description 5
- 239000007795 chemical reaction product Substances 0.000 description 5
- 230000007423 decrease Effects 0.000 description 5
- 239000000539 dimer Substances 0.000 description 5
- 230000002068 genetic effect Effects 0.000 description 5
- 238000000338 in vitro Methods 0.000 description 5
- 230000003993 interaction Effects 0.000 description 5
- 230000003426 interchromosomal effect Effects 0.000 description 5
- 230000005291 magnetic effect Effects 0.000 description 5
- 230000007246 mechanism Effects 0.000 description 5
- 230000008569 process Effects 0.000 description 5
- 238000000746 purification Methods 0.000 description 5
- 239000011541 reaction mixture Substances 0.000 description 5
- 101150031434 tnp2 gene Proteins 0.000 description 5
- 238000012546 transfer Methods 0.000 description 5
- 239000004971 Cross linker Substances 0.000 description 4
- 102000004190 Enzymes Human genes 0.000 description 4
- 108090000790 Enzymes Proteins 0.000 description 4
- 102000012330 Integrases Human genes 0.000 description 4
- 239000011543 agarose gel Substances 0.000 description 4
- 238000000246 agarose gel electrophoresis Methods 0.000 description 4
- 125000000539 amino acid group Chemical group 0.000 description 4
- 239000012472 biological sample Substances 0.000 description 4
- 210000001124 body fluid Anatomy 0.000 description 4
- 239000010839 body fluid Substances 0.000 description 4
- 235000018417 cysteine Nutrition 0.000 description 4
- XUJNEKJLAYXESH-UHFFFAOYSA-N cysteine Natural products SCC(N)C(O)=O XUJNEKJLAYXESH-UHFFFAOYSA-N 0.000 description 4
- 238000002474 experimental method Methods 0.000 description 4
- 239000012530 fluid Substances 0.000 description 4
- 102000054766 genetic haplotypes Human genes 0.000 description 4
- 230000001404 mediated effect Effects 0.000 description 4
- 108010009127 mu transposase Proteins 0.000 description 4
- 238000005457 optimization Methods 0.000 description 4
- 229920000642 polymer Polymers 0.000 description 4
- 108090000765 processed proteins & peptides Proteins 0.000 description 4
- 238000012545 processing Methods 0.000 description 4
- 238000000926 separation method Methods 0.000 description 4
- WBHQBSYUUJJSRZ-UHFFFAOYSA-M sodium bisulfate Chemical compound [Na+].OS([O-])(=O)=O WBHQBSYUUJJSRZ-UHFFFAOYSA-M 0.000 description 4
- 229910000342 sodium bisulfate Inorganic materials 0.000 description 4
- 238000006467 substitution reaction Methods 0.000 description 4
- 239000000758 substrate Substances 0.000 description 4
- 238000012360 testing method Methods 0.000 description 4
- -1 2-amino-dA Chemical compound 0.000 description 3
- LRSASMSXMSNRBT-UHFFFAOYSA-N 5-methylcytosine Chemical compound CC1=CNC(=O)N=C1N LRSASMSXMSNRBT-UHFFFAOYSA-N 0.000 description 3
- 102000006947 Histones Human genes 0.000 description 3
- 239000004594 Masterbatch (MB) Substances 0.000 description 3
- 108010092799 RNA-directed DNA polymerase Proteins 0.000 description 3
- 150000001412 amines Chemical class 0.000 description 3
- 235000001014 amino acid Nutrition 0.000 description 3
- 108010027090 biotin-streptavidin complex Proteins 0.000 description 3
- 125000000151 cysteine group Chemical group N[C@@H](CS)C(=O)* 0.000 description 3
- 238000011161 development Methods 0.000 description 3
- 230000018109 developmental process Effects 0.000 description 3
- WBZKQQHYRPRKNJ-UHFFFAOYSA-L disulfite Chemical compound [O-]S(=O)S([O-])(=O)=O WBZKQQHYRPRKNJ-UHFFFAOYSA-L 0.000 description 3
- 238000011049 filling Methods 0.000 description 3
- 230000000670 limiting effect Effects 0.000 description 3
- 238000013507 mapping Methods 0.000 description 3
- 239000000463 material Substances 0.000 description 3
- 108020004999 messenger RNA Proteins 0.000 description 3
- 238000012986 modification Methods 0.000 description 3
- 239000002105 nanoparticle Substances 0.000 description 3
- 210000003463 organelle Anatomy 0.000 description 3
- 102000040430 polynucleotide Human genes 0.000 description 3
- 108091033319 polynucleotide Proteins 0.000 description 3
- 239000002157 polynucleotide Substances 0.000 description 3
- 229920001184 polypeptide Polymers 0.000 description 3
- 102000004196 processed proteins & peptides Human genes 0.000 description 3
- 108091008146 restriction endonucleases Proteins 0.000 description 3
- 238000001542 size-exclusion chromatography Methods 0.000 description 3
- 238000000527 sonication Methods 0.000 description 3
- 210000001519 tissue Anatomy 0.000 description 3
- RYVNIFSIEDRLSJ-UHFFFAOYSA-N 5-(hydroxymethyl)cytosine Chemical compound NC=1NC(=O)N=CC=1CO RYVNIFSIEDRLSJ-UHFFFAOYSA-N 0.000 description 2
- YBJHBAHKTGYVGT-ZXFLCMHBSA-N 5-[(3ar,4r,6as)-2-oxo-1,3,3a,4,6,6a-hexahydrothieno[3,4-d]imidazol-4-yl]pentanoic acid Chemical compound N1C(=O)N[C@H]2[C@@H](CCCCC(=O)O)SC[C@H]21 YBJHBAHKTGYVGT-ZXFLCMHBSA-N 0.000 description 2
- 108700028369 Alleles Proteins 0.000 description 2
- 108091093088 Amplicon Proteins 0.000 description 2
- 102000012410 DNA Ligases Human genes 0.000 description 2
- 108010061982 DNA Ligases Proteins 0.000 description 2
- 101100310856 Drosophila melanogaster spri gene Proteins 0.000 description 2
- 108091092584 GDNA Proteins 0.000 description 2
- 241000713869 Moloney murine leukemia virus Species 0.000 description 2
- 108010047956 Nucleosomes Proteins 0.000 description 2
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 2
- 239000012506 Sephacryl® Substances 0.000 description 2
- VYPSYNLAJGMNEJ-UHFFFAOYSA-N Silicium dioxide Chemical compound O=[Si]=O VYPSYNLAJGMNEJ-UHFFFAOYSA-N 0.000 description 2
- 108020004682 Single-Stranded DNA Proteins 0.000 description 2
- GWEVSGVZZGPLCZ-UHFFFAOYSA-N Titan oxide Chemical compound O=[Ti]=O GWEVSGVZZGPLCZ-UHFFFAOYSA-N 0.000 description 2
- 125000003275 alpha amino acid group Chemical group 0.000 description 2
- 150000001413 amino acids Chemical class 0.000 description 2
- 125000003277 amino group Chemical group 0.000 description 2
- 230000001580 bacterial effect Effects 0.000 description 2
- 230000003139 buffering effect Effects 0.000 description 2
- 239000006227 byproduct Substances 0.000 description 2
- 201000011510 cancer Diseases 0.000 description 2
- 230000015556 catabolic process Effects 0.000 description 2
- 108091092356 cellular DNA Proteins 0.000 description 2
- 230000008859 change Effects 0.000 description 2
- 239000003153 chemical reaction reagent Substances 0.000 description 2
- 238000006731 degradation reaction Methods 0.000 description 2
- 238000004925 denaturation Methods 0.000 description 2
- 230000036425 denaturation Effects 0.000 description 2
- 238000010790 dilution Methods 0.000 description 2
- 239000012895 dilution Substances 0.000 description 2
- 229940079593 drug Drugs 0.000 description 2
- 239000003814 drug Substances 0.000 description 2
- 238000006911 enzymatic reaction Methods 0.000 description 2
- 238000001976 enzyme digestion Methods 0.000 description 2
- QAOWNCQODCNURD-UHFFFAOYSA-M hydrogensulfate Chemical compound OS([O-])(=O)=O QAOWNCQODCNURD-UHFFFAOYSA-M 0.000 description 2
- 230000003100 immobilizing effect Effects 0.000 description 2
- 230000006872 improvement Effects 0.000 description 2
- 230000009319 interchromosomal translocation Effects 0.000 description 2
- 230000014759 maintenance of location Effects 0.000 description 2
- 229910052751 metal Inorganic materials 0.000 description 2
- 239000002184 metal Substances 0.000 description 2
- 150000002739 metals Chemical class 0.000 description 2
- 230000004048 modification Effects 0.000 description 2
- 210000001623 nucleosome Anatomy 0.000 description 2
- 230000003169 placental effect Effects 0.000 description 2
- 239000013615 primer Substances 0.000 description 2
- 239000002987 primer (paints) Substances 0.000 description 2
- 238000011084 recovery Methods 0.000 description 2
- 230000001105 regulatory effect Effects 0.000 description 2
- 230000008439 repair process Effects 0.000 description 2
- 230000000717 retained effect Effects 0.000 description 2
- 230000001177 retroviral effect Effects 0.000 description 2
- 210000002966 serum Anatomy 0.000 description 2
- 239000011550 stock solution Substances 0.000 description 2
- JJAHTWIKCUJRDK-UHFFFAOYSA-N succinimidyl 4-(N-maleimidomethyl)cyclohexane-1-carboxylate Chemical compound C1CC(CN2C(C=CC2=O)=O)CCC1C(=O)ON1C(=O)CCC1=O JJAHTWIKCUJRDK-UHFFFAOYSA-N 0.000 description 2
- 238000001847 surface plasmon resonance imaging Methods 0.000 description 2
- 239000000725 suspension Substances 0.000 description 2
- 230000009466 transformation Effects 0.000 description 2
- VYMHBQQZUYHXSS-UHFFFAOYSA-N 2-(3h-dithiol-3-yl)pyridine Chemical group C1=CSSC1C1=CC=CC=N1 VYMHBQQZUYHXSS-UHFFFAOYSA-N 0.000 description 1
- JLBJTVDPSNHSKJ-UHFFFAOYSA-N 4-Methylstyrene Chemical compound CC1=CC=C(C=C)C=C1 JLBJTVDPSNHSKJ-UHFFFAOYSA-N 0.000 description 1
- ZLAQATDNGLKIEV-UHFFFAOYSA-N 5-methyl-2-sulfanylidene-1h-pyrimidin-4-one Chemical compound CC1=CNC(=S)NC1=O ZLAQATDNGLKIEV-UHFFFAOYSA-N 0.000 description 1
- TVICROIWXBFQEL-UHFFFAOYSA-N 6-(ethylamino)-1h-pyrimidin-2-one Chemical compound CCNC1=CC=NC(=O)N1 TVICROIWXBFQEL-UHFFFAOYSA-N 0.000 description 1
- 229920000936 Agarose Polymers 0.000 description 1
- 241000589158 Agrobacterium Species 0.000 description 1
- 241000193830 Bacillus <bacterium> Species 0.000 description 1
- 241000894006 Bacteria Species 0.000 description 1
- 241000589968 Borrelia Species 0.000 description 1
- 241000244203 Caenorhabditis elegans Species 0.000 description 1
- 102100033620 Calponin-1 Human genes 0.000 description 1
- OKTJSMMVPCPJKN-UHFFFAOYSA-N Carbon Chemical compound [C] OKTJSMMVPCPJKN-UHFFFAOYSA-N 0.000 description 1
- 102000014914 Carrier Proteins Human genes 0.000 description 1
- 241000282693 Cercopithecidae Species 0.000 description 1
- 241000606161 Chlamydia Species 0.000 description 1
- 108020004998 Chloroplast DNA Proteins 0.000 description 1
- 241000193403 Clostridium Species 0.000 description 1
- 208000035473 Communicable disease Diseases 0.000 description 1
- 108010001237 Cytochrome P-450 CYP2D6 Proteins 0.000 description 1
- 102100021704 Cytochrome P450 2D6 Human genes 0.000 description 1
- YTBSYETUWUMLBZ-QWWZWVQMSA-N D-threose Chemical compound OC[C@@H](O)[C@H](O)C=O YTBSYETUWUMLBZ-QWWZWVQMSA-N 0.000 description 1
- 230000007118 DNA alkylation Effects 0.000 description 1
- 230000005778 DNA damage Effects 0.000 description 1
- 231100000277 DNA damage Toxicity 0.000 description 1
- 230000007067 DNA methylation Effects 0.000 description 1
- 229920002307 Dextran Polymers 0.000 description 1
- 241000255581 Drosophila <fruit fly, genus> Species 0.000 description 1
- 241000196324 Embryophyta Species 0.000 description 1
- 241000588698 Erwinia Species 0.000 description 1
- 241000588722 Escherichia Species 0.000 description 1
- 241000233866 Fungi Species 0.000 description 1
- 208000034826 Genetic Predisposition to Disease Diseases 0.000 description 1
- 208000031886 HIV Infections Diseases 0.000 description 1
- 241000589989 Helicobacter Species 0.000 description 1
- 241000405147 Hermes Species 0.000 description 1
- 241000238631 Hexapoda Species 0.000 description 1
- 241000282412 Homo Species 0.000 description 1
- 101000945318 Homo sapiens Calponin-1 Proteins 0.000 description 1
- 101000652736 Homo sapiens Transgelin Proteins 0.000 description 1
- 101000690100 Homo sapiens U1 small nuclear ribonucleoprotein 70 kDa Proteins 0.000 description 1
- 241000713772 Human immunodeficiency virus 1 Species 0.000 description 1
- 241000713340 Human immunodeficiency virus 2 Species 0.000 description 1
- WHUUTDBJXJRKMK-VKHMYHEASA-N L-glutamic acid Chemical compound OC(=O)[C@@H](N)CCC(O)=O WHUUTDBJXJRKMK-VKHMYHEASA-N 0.000 description 1
- 241000589248 Legionella Species 0.000 description 1
- 208000007764 Legionnaires' Disease Diseases 0.000 description 1
- 102000003960 Ligases Human genes 0.000 description 1
- 108090000364 Ligases Proteins 0.000 description 1
- 208000016604 Lyme disease Diseases 0.000 description 1
- PEEHTFAAVSWFBL-UHFFFAOYSA-N Maleimide Chemical compound O=C1NC(=O)C=C1 PEEHTFAAVSWFBL-UHFFFAOYSA-N 0.000 description 1
- 241000124008 Mammalia Species 0.000 description 1
- 241001465754 Metazoa Species 0.000 description 1
- 108020005196 Mitochondrial DNA Proteins 0.000 description 1
- YNAVUWVOSKDBBP-UHFFFAOYSA-N Morpholine Natural products C1COCCN1 YNAVUWVOSKDBBP-UHFFFAOYSA-N 0.000 description 1
- 241000699670 Mus sp. Species 0.000 description 1
- 241000186359 Mycobacterium Species 0.000 description 1
- 241000204031 Mycoplasma Species 0.000 description 1
- 241000588653 Neisseria Species 0.000 description 1
- 241000244206 Nematoda Species 0.000 description 1
- 102000007999 Nuclear Proteins Human genes 0.000 description 1
- 108010089610 Nuclear Proteins Proteins 0.000 description 1
- 239000004677 Nylon Substances 0.000 description 1
- 240000007019 Oxalis corniculata Species 0.000 description 1
- 102000035195 Peptidases Human genes 0.000 description 1
- 108091005804 Peptidases Proteins 0.000 description 1
- 101100029173 Phaeosphaeria nodorum (strain SN15 / ATCC MYA-4574 / FGSC 10173) SNP2 gene Proteins 0.000 description 1
- 108020005120 Plant DNA Proteins 0.000 description 1
- 208000002151 Pleural effusion Diseases 0.000 description 1
- 208000020584 Polyploidy Diseases 0.000 description 1
- 239000004793 Polystyrene Substances 0.000 description 1
- 206010036790 Productive cough Diseases 0.000 description 1
- 241000589516 Pseudomonas Species 0.000 description 1
- 241000910071 Pyrobaculum filamentous virus 1 Species 0.000 description 1
- 241000700157 Rattus norvegicus Species 0.000 description 1
- 241000589180 Rhizobium Species 0.000 description 1
- 101100094821 Saccharomyces cerevisiae (strain ATCC 204508 / S288c) SMX2 gene Proteins 0.000 description 1
- 241000607142 Salmonella Species 0.000 description 1
- 229920002684 Sepharose Polymers 0.000 description 1
- 241000607720 Serratia Species 0.000 description 1
- DWAQJAXMDSEUJJ-UHFFFAOYSA-M Sodium bisulfite Chemical compound [Na+].OS([O-])=O DWAQJAXMDSEUJJ-UHFFFAOYSA-M 0.000 description 1
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 1
- 241000191940 Staphylococcus Species 0.000 description 1
- 241000191967 Staphylococcus aureus Species 0.000 description 1
- 241000194017 Streptococcus Species 0.000 description 1
- 241000187747 Streptomyces Species 0.000 description 1
- 239000004809 Teflon Substances 0.000 description 1
- 229920006362 Teflon® Polymers 0.000 description 1
- RYYWUUFWQRZTIU-UHFFFAOYSA-N Thiophosphoric acid Chemical group OP(O)(S)=O RYYWUUFWQRZTIU-UHFFFAOYSA-N 0.000 description 1
- 150000001218 Thorium Chemical class 0.000 description 1
- 241000589886 Treponema Species 0.000 description 1
- 102100024121 U1 small nuclear ribonucleoprotein 70 kDa Human genes 0.000 description 1
- 230000005856 abnormality Effects 0.000 description 1
- 230000009471 action Effects 0.000 description 1
- 210000004381 amniotic fluid Anatomy 0.000 description 1
- 238000000137 annealing Methods 0.000 description 1
- 230000002583 anti-histone Effects 0.000 description 1
- 238000003491 array Methods 0.000 description 1
- 125000000852 azido group Chemical group *N=[N+]=[N-] 0.000 description 1
- XMQFTWRPUQYINF-UHFFFAOYSA-N bensulfuron-methyl Chemical compound COC(=O)C1=CC=CC=C1CS(=O)(=O)NC(=O)NC1=NC(OC)=CC(OC)=N1 XMQFTWRPUQYINF-UHFFFAOYSA-N 0.000 description 1
- 108091008324 binding proteins Proteins 0.000 description 1
- 230000033228 biological regulation Effects 0.000 description 1
- 238000006664 bond formation reaction Methods 0.000 description 1
- 239000004202 carbamide Substances 0.000 description 1
- 150000001720 carbohydrates Chemical group 0.000 description 1
- 230000024245 cell differentiation Effects 0.000 description 1
- 239000013592 cell lysate Substances 0.000 description 1
- 210000000170 cell membrane Anatomy 0.000 description 1
- 230000019522 cellular metabolic process Effects 0.000 description 1
- 229920002678 cellulose Polymers 0.000 description 1
- 239000001913 cellulose Substances 0.000 description 1
- 238000005119 centrifugation Methods 0.000 description 1
- 239000000919 ceramic Substances 0.000 description 1
- 210000001175 cerebrospinal fluid Anatomy 0.000 description 1
- 238000002144 chemical decomposition reaction Methods 0.000 description 1
- 238000003776 cleavage reaction Methods 0.000 description 1
- 239000003086 colorant Substances 0.000 description 1
- 238000004440 column chromatography Methods 0.000 description 1
- 239000012141 concentrate Substances 0.000 description 1
- 230000001268 conjugating effect Effects 0.000 description 1
- 238000011109 contamination Methods 0.000 description 1
- 230000006378 damage Effects 0.000 description 1
- 230000006837 decompression Effects 0.000 description 1
- 239000005546 dideoxynucleotide Substances 0.000 description 1
- 230000004069 differentiation Effects 0.000 description 1
- 239000012470 diluted sample Substances 0.000 description 1
- 201000010099 disease Diseases 0.000 description 1
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 1
- 238000005516 engineering process Methods 0.000 description 1
- 230000002255 enzymatic effect Effects 0.000 description 1
- 210000003527 eukaryotic cell Anatomy 0.000 description 1
- 238000011156 evaluation Methods 0.000 description 1
- 238000001914 filtration Methods 0.000 description 1
- 125000000524 functional group Chemical group 0.000 description 1
- 239000000499 gel Substances 0.000 description 1
- 238000012239 gene modification Methods 0.000 description 1
- 230000005017 genetic modification Effects 0.000 description 1
- 235000013617 genetically modified food Nutrition 0.000 description 1
- 239000011521 glass Substances 0.000 description 1
- 229930195712 glutamate Natural products 0.000 description 1
- 150000004676 glycans Chemical class 0.000 description 1
- 239000010439 graphite Substances 0.000 description 1
- 229910002804 graphite Inorganic materials 0.000 description 1
- 150000003278 haem Chemical class 0.000 description 1
- 108091008039 hormone receptors Proteins 0.000 description 1
- 235000020256 human milk Nutrition 0.000 description 1
- 210000004251 human milk Anatomy 0.000 description 1
- 230000007062 hydrolysis Effects 0.000 description 1
- 238000006460 hydrolysis reaction Methods 0.000 description 1
- 239000012535 impurity Substances 0.000 description 1
- 238000010348 incorporation Methods 0.000 description 1
- 238000011534 incubation Methods 0.000 description 1
- 230000010354 integration Effects 0.000 description 1
- 238000002955 isolation Methods 0.000 description 1
- 239000004816 latex Substances 0.000 description 1
- 229920000126 latex Polymers 0.000 description 1
- 239000003446 ligand Substances 0.000 description 1
- 238000011068 loading method Methods 0.000 description 1
- 210000002751 lymph Anatomy 0.000 description 1
- 239000006166 lysate Substances 0.000 description 1
- 238000012423 maintenance Methods 0.000 description 1
- 102000016470 mariner transposase Human genes 0.000 description 1
- 108060004631 mariner transposase Proteins 0.000 description 1
- 239000012528 membrane Substances 0.000 description 1
- 125000002496 methyl group Chemical group [H]C([H])([H])* 0.000 description 1
- 239000000693 micelle Substances 0.000 description 1
- 244000005700 microbiome Species 0.000 description 1
- 210000003470 mitochondria Anatomy 0.000 description 1
- 239000013642 negative control Substances 0.000 description 1
- 210000004940 nucleus Anatomy 0.000 description 1
- 229920001778 nylon Polymers 0.000 description 1
- 210000000056 organ Anatomy 0.000 description 1
- 230000003647 oxidation Effects 0.000 description 1
- 238000007254 oxidation reaction Methods 0.000 description 1
- 230000005298 paramagnetic effect Effects 0.000 description 1
- 239000002907 paramagnetic material Substances 0.000 description 1
- 244000045947 parasite Species 0.000 description 1
- 230000007170 pathology Effects 0.000 description 1
- 239000013610 patient sample Substances 0.000 description 1
- 125000002467 phosphate group Chemical group [H]OP(=O)(O[H])O[*] 0.000 description 1
- 239000004033 plastic Substances 0.000 description 1
- 229920000058 polyacrylate Polymers 0.000 description 1
- 229920001282 polysaccharide Polymers 0.000 description 1
- 239000005017 polysaccharide Substances 0.000 description 1
- 229920002223 polystyrene Polymers 0.000 description 1
- 230000001376 precipitating effect Effects 0.000 description 1
- 210000001236 prokaryotic cell Anatomy 0.000 description 1
- 230000002035 prolonged effect Effects 0.000 description 1
- 235000019833 protease Nutrition 0.000 description 1
- 108020003175 receptors Proteins 0.000 description 1
- 230000009467 reduction Effects 0.000 description 1
- 230000002829 reductive effect Effects 0.000 description 1
- 230000003252 repetitive effect Effects 0.000 description 1
- 230000004044 response Effects 0.000 description 1
- 238000003757 reverse transcription PCR Methods 0.000 description 1
- 230000002441 reversible effect Effects 0.000 description 1
- 238000012552 review Methods 0.000 description 1
- 210000003296 saliva Anatomy 0.000 description 1
- 150000003839 salts Chemical class 0.000 description 1
- 238000005070 sampling Methods 0.000 description 1
- 230000007017 scission Effects 0.000 description 1
- 238000004062 sedimentation Methods 0.000 description 1
- 210000000582 semen Anatomy 0.000 description 1
- 238000010008 shearing Methods 0.000 description 1
- 239000000377 silicon dioxide Substances 0.000 description 1
- 239000011780 sodium chloride Substances 0.000 description 1
- 235000010267 sodium hydrogen sulphite Nutrition 0.000 description 1
- 210000003802 sputum Anatomy 0.000 description 1
- 208000024794 sputum Diseases 0.000 description 1
- 238000010561 standard procedure Methods 0.000 description 1
- 230000000638 stimulation Effects 0.000 description 1
- 238000012916 structural analysis Methods 0.000 description 1
- 230000000946 synaptic effect Effects 0.000 description 1
- 230000008685 targeting Effects 0.000 description 1
- 210000001138 tear Anatomy 0.000 description 1
- 125000004149 thio group Chemical group *S* 0.000 description 1
- 125000003396 thiol group Chemical group [H]S* 0.000 description 1
- 239000004408 titanium dioxide Substances 0.000 description 1
- 238000004448 titration Methods 0.000 description 1
- 230000005945 translocation Effects 0.000 description 1
- 230000013819 transposition, DNA-mediated Effects 0.000 description 1
- 210000004881 tumor cell Anatomy 0.000 description 1
- 241001430294 unidentified retrovirus Species 0.000 description 1
- 210000002700 urine Anatomy 0.000 description 1
- 238000005406 washing Methods 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/10—Processes for the isolation, preparation or purification of DNA or RNA
- C12N15/1034—Isolating an individual clone by screening libraries
- C12N15/1065—Preparation or screening of tagged libraries, e.g. tagged microorganisms by STM-mutagenesis, tagged polynucleotides, gene tags
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/10—Processes for the isolation, preparation or purification of DNA or RNA
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/10—Processes for the isolation, preparation or purification of DNA or RNA
- C12N15/1034—Isolating an individual clone by screening libraries
- C12N15/1082—Preparation or screening gene libraries by chromosomal integration of polynucleotide sequences, HR-, site-specific-recombination, transposons, viral vectors
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/10—Processes for the isolation, preparation or purification of DNA or RNA
- C12N15/1034—Isolating an individual clone by screening libraries
- C12N15/1093—General methods of preparing gene libraries, not provided for in other subgroups
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6806—Preparing nucleic acids for analysis, e.g. for polymerase chain reaction [PCR] assay
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6813—Hybridisation assays
- C12Q1/6834—Enzymatic or biochemical coupling of nucleic acids to a solid phase
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6869—Methods for sequencing
- C12Q1/6874—Methods for sequencing involving nucleic acid arrays, e.g. sequencing by hybridisation
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6876—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes
-
- C—CHEMISTRY; METALLURGY
- C40—COMBINATORIAL TECHNOLOGY
- C40B—COMBINATORIAL CHEMISTRY; LIBRARIES, e.g. CHEMICAL LIBRARIES
- C40B50/00—Methods of creating libraries, e.g. combinatorial synthesis
- C40B50/06—Biochemical methods, e.g. using enzymes or whole viable microorganisms
-
- C—CHEMISTRY; METALLURGY
- C40—COMBINATORIAL TECHNOLOGY
- C40B—COMBINATORIAL CHEMISTRY; LIBRARIES, e.g. CHEMICAL LIBRARIES
- C40B70/00—Tags or labels specially adapted for combinatorial chemistry or libraries, e.g. fluorescent tags or bar codes
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2525/00—Reactions involving modified oligonucleotides, nucleic acids, or nucleotides
- C12Q2525/10—Modifications characterised by
- C12Q2525/191—Modifications characterised by incorporating an adaptor
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2563/00—Nucleic acid detection characterized by the use of physical, structural and functional properties
- C12Q2563/179—Nucleic acid detection characterized by the use of physical, structural and functional properties the label being a nucleic acid
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2565/00—Nucleic acid analysis characterised by mode or means of detection
- C12Q2565/50—Detection characterised by immobilisation to a surface
- C12Q2565/514—Detection characterised by immobilisation to a surface characterised by the use of the arrayed oligonucleotides as identifier tags, e.g. universal addressable array, anti-tag or tag complement array
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/154—Methylation markers
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/156—Polymorphic or mutational markers
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/172—Haplotypes
Landscapes
- Chemical & Material Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Health & Medical Sciences (AREA)
- Organic Chemistry (AREA)
- Engineering & Computer Science (AREA)
- Genetics & Genomics (AREA)
- Zoology (AREA)
- Wood Science & Technology (AREA)
- Biotechnology (AREA)
- General Engineering & Computer Science (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Biochemistry (AREA)
- Molecular Biology (AREA)
- Microbiology (AREA)
- Biomedical Technology (AREA)
- General Health & Medical Sciences (AREA)
- Biophysics (AREA)
- Physics & Mathematics (AREA)
- Analytical Chemistry (AREA)
- Plant Pathology (AREA)
- Crystallography & Structural Chemistry (AREA)
- Immunology (AREA)
- Bioinformatics & Computational Biology (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Medicinal Chemistry (AREA)
- General Chemical & Material Sciences (AREA)
- Virology (AREA)
- Measuring Or Testing Involving Enzymes Or Micro-Organisms (AREA)
- Immobilizing And Processing Of Enzymes And Microorganisms (AREA)
- Glass Compositions (AREA)
- Inorganic Insulating Materials (AREA)
- Transition And Organic Metals Composition Catalysts For Addition Polymerization (AREA)
Abstract
Изобретение относится к области биотехнологии. Предложена композиция для получения библиотеки. Композиция содержит множество клонально индексированных твердых носителей, где каждый из твердых носителей иммобилизует на себе множество олигонуклеотидов. Каждый из множества иммобилизованных олигонуклеотидов содержит комплементарную последовательность для захвата для иммобилизации на твердом носителе ассоциированного c нуклеиновой кислотой-мишенью транспозомного комплекса, первую последовательность со штрих–кодом и сайт связывания с праймером. Причем первая последовательность со штрих–кодом с каждого твердого носителя отличается от всех первых последовательностей со штрих–кодом с других твердых носителей, все из иммобилизованных олигонуклеотидов содержат одну и ту же комплементарную последовательность для захвата и все из иммобилизованных олигонуклеотидов содержат одну и ту же первую последовательность со штрих–кодом. Транспозомный комплекс содержит связанный с транспозазой транспозон. Изобретение обеспечивает получение информации о сцеплении генов, о фазировании и определение статуса метилирования нуклеиновой кислоты-мишени. 14 з.п. ф-лы, 21 пр., 4 табл., 83 ил.
Description
Родственнные заявки
В настоящей заявке испрашивается приоритет предварительной заявки на патент США Nо: 62/065544, поданной 17 октября, 2014, и предварительной заявки на патент США Nо: 62/157396, поданной 5 мая, 2015, которые во всей своей полноте вводятся в настоящее описание посредством ссылки.
Область, к которой относится изобретение
Варианты настоящего изобретения относятся к секвенированию нуклеиновых кислот. В частности, описанные здесь варианты способов и композиций относятся к получению матриц на основе нуклеиновых кислот и к получению данных для этих последовательностей.
Предшествующий уровень техники
Детектирование специфических последовательностей нуклеиновых кислот, присутствующих в биологическом образце, применяется, например, как способ идентификации и классификации микроорганизмов, диагностики инфекционных заболеваний, детектирования и характеризации генетических аномалий, идентификации генетических модификаций, ассоциированных с развитием рака, исследования генетической восприимчивости к развитию заболевания и оценки ответа на различные типы лечения. Общим методом детектирования специфических последовательностей нуклеиновых кислот в биологическом образце является секвенирование нуклеиновых кислот.
Методика секвенирования нуклеиновых кислот была разработана, главным образом, на основе методов химического разложения, применяемых Mэксэмом и Гилбертом, и метода удлинения цепи, используемого Сэнгером. В настоящее время применяется несколько методов секвенирования, которые позволяют осуществлять параллельный процессинг всех нуклеиновых кислот в одном раунде секвенирования. При этом, информация, получаемая после проведения одного раунда секвенирования, может быть слишком объемной.
Описание сущности изобретения
В одном из аспектов настоящего изобретения описаны способы получения библиотеки фрагментов ДНК нуклеиновой кислоты-мишени со штрих-кодами. Эти способы включают контактирование нуклеиновой кислоты-мишени с множеством транспозомных комплексов, каждый из которых включает транспозоны и транспозазы, где транспозоны содержат перенесенные цепи и неперенесенные цепи. По меньшей мере один из транспозонов транспозомного комплекса включает последовательность адаптера, способную гибридизоваться с комплементарной последовательностью для захвата. Нуклеиновую кислоту-мишень фрагментируют с образованием множества фрагментов, а затем множество перенесенных цепей встраивают в 5'-конец по меньшей мере одной цепи фрагментов с сохранением сцепления нуклеиновой кислоты-мишени. Множество фрагментов нуклеиновой кислоты-мишени подвергают контактированию с множеством твердых носителей, каждый из которых содержит множество иммобилизованных олигонуклеотидов, где каждый из этих олигонуклеотидов включает комплементарную последовательность для захвата и первую последовательность со штрих-кодом, где первая последовательность со штрих-кодом, присутствующая на каждом твердом носителе из множества твердых носителей, отличается от первой последовательности со штрих-кодом, присутствующей на других твердых носителях из множества твердых носителей. Информацию о последовательности со штрих-кодом переносят во фрагменты нуклеиновой кислоты-мишени, в результате чего получают иммобилизованную библиотеку двухцепочечных фрагментов, где по меньшей мере одна цепь помечена у 5'-конца первым штрих-кодом так, чтобы по меньшей мере два фрагмента одной и той же нуклеиновой кислоты-мишени получали идентичную информацию по штрих-коду.
В одном из аспектов изобретения описаны способы получения информации о сцеплении последовательности нуклеиновой кислоты-мишени. Эти способы включают контактирование нуклеиновой кислоты-мишени с множеством транспозомных комплексов, каждый из которых включает транспозоны и транспозазы, где транспозоны содержат перенесенные цепи и неперенесенные цепи, в которых по меньшей мере один из транспозонов транспозомного комплекса включает последовательность адаптера, способную гибридизоваться с комплементарной последовательностью для захвата. Нуклеиновую кислоту-мишень фрагментируют с образованием множества фрагментов, а затем множество перенесенных цепей встраивают во множество фрагментов с сохранением сцепления нуклеиновой кислоты-мишени. Множество фрагментов нуклеиновой кислоты-мишени подвергают контактированию с множеством твердых носителей. Каждый из этих твердых носителей содержит множество иммобилизованных олигонуклеотидов, которые включают комплементарную последовательность для захвата и первую последовательность со штрих-кодом, где первая последовательность со штрих-кодом, присутствующая на каждом твердом носителе из множества твердых носителей, отличается от первой последовательности со штрих-кодом, присутствующей на других твердых носителях из множества твердых носителей. Информацию о последовательности со штрих-кодом переносят во фрагменты нуклеиновой кислоты-мишени, так, чтобы по меньшей мере два фрагмента одной и той же нуклеиновой кислоты-мишени получали идентичную информацию по штрих-коду. Затем определяют последовательность фрагментов нуклеиновой кислоты-мишени и последовательностей со штрих-кодом. Информацию о сцеплении нуклеиновой кислоты-мишени определяют путем идентификации последовательностей со штрих-кодом. В некоторых вариантах осуществления изобретения, транспозазы транспозомных комплексов удаляют после транспозиции и последующей гибридизации последовательностей адаптеров транспозона с комплементарной последовательностью для захвата. В некоторых вариантах осуществления изобретения, транспозазы удаляют путем ДСН-обработки. В некоторых вариантах осуществления изобретения, транспозазы удаляют путем обработки протеиназой.
В одном из аспектов изобретения описаны способы получения одновременной информации о фазах и статусе метилирования последовательности нуклеиновой кислоты-мишени. Эти способы включают контактирование нуклеиновой кислоты-мишени с множеством транспозомных комплексов, каждый из которых включает транспозоны и транспозазы, где транспозоны содержат перенесенные цепи и неперенесенные цепи, в которых по меньшей мере один из транспозонов транспозомного комплекса включает последовательность адаптера, способную гибридизоваться с комплементарной последовательностью для захвата. Нуклеиновую кислоту-мишень фрагментируют с образованием множества фрагментов, а затем множество перенесенных цепей встраивают во фрагменты нуклеиновой кислоты-мишени с сохранением сцепления нуклеиновой кислоты-мишени. Множество фрагментов нуклеиновой кислоты-мишени подвергают контактированию с множеством твердых носителей, каждый из которых содержит множество иммобилизованных олигонуклеотидов, где каждый из этих олигонуклеотидов включает комплементарную последовательность для захвата и первую последовательность со штрих-кодом, и где первая последовательность со штрих-кодом, присутствующая на каждом твердом носителе из множества твердых носителей, отличается от первой последовательности со штрих-кодом, присутствующей на других твердых носителях из множества твердых носителей. Информацию о последовательности со штрих-кодом переносят во фрагменты нуклеиновой кислоты-мишени, так, чтобы по меньшей мере два фрагмента одной и той же нуклеиновой кислоты-мишени получали идентичную информацию по штрих-коду. Затем фрагменты нуклеиновой кислоты-мишени, содержащие штрих-коды, подвергают обработке бисульфитом, в результате чего получают обработанные бисульфитом фрагменты нуклеиновой кислоты-мишени, содержащие штрих-коды. После этого определяют последовательность обработанных бисульфитом фрагментов нуклеиновой кислоты-мишени и последовательности со штрих-кодом. Информацию о сцеплении нуклеиновой кислоты-мишени определяют путем идентификации последовательностей со штрих-кодом.
В одном из аспектов изобретения описаны способы получения иммобилизованной библиотеки меченных фрагментов ДНК. Эти способы включают получение множества твердых носителей, имеющих транспозомные комплексы, иммобилизованные на этих носителях, где указанные транспозомные комплексы являются мультимерными, а транспозомные мономерные единицы одного и того же транспозомного комплекса являются сцепленными друг с другом, и где указанные транспозомные мономерные единицы содержат транспозазу, связанную с первым полинуклеотидом, где указанный первый полинуклеотид включает (i) 3'-часть, содержащую концевую последовательность транспозона, и (ii) первый адаптер, содержащий первый штрих-код. ДНК-мишень наносят на множество твердых носителей в условиях, при которых ДНК-мишень фрагментируется транспозомными комплексами, а 3'-концевую последовательность транспозона первого полинуклеотида переносят в 5'-конец по меньшей мере одной цепи фрагментов, в результате чего получают иммобилизованную библиотеку двухцепочечных фрагментов, в которых по меньшей мере одна цепь помечена у 5'-конца первым штрих-кодом.
В одном из аспектов изобретения описаны способы получения секвенирующей библиотеки для определения статуса метилирования нуклеиновой кислоты-мишени. Эти способы включают фрагментирование нуклеиновой кислоты-мишени с получением двух или более фрагментов. Первую общую последовательность адаптера вводят в 5'-конец фрагментов нуклеиновой кислоты-мишени, где указанная последовательность адаптера содержит первую последовательность, связывающуюся с праймером, и аффинную группу, где указанная аффинная группа является одним из членов связывающейся пары. Фрагмент нуклеиновой кислоты-мишени подвергают денатурации. Затем, фрагмент нуклеиновой кислоты-мишени иммобилизуют на твердом носителе, где указанный твердый носитель включает другой член связывающейся пары, и где иммобилизация нуклеиновой кислоты-мишени представляет собой связывание связывающейся пары. Иммобилизованные фрагменты нуклеиновой кислоты-мишени подвергают обработке дисульфитом. Вторую общую последовательность адаптера включают в иммобилизованные фрагменты нуклеиновой кислоты-мишени, обработанные бисульфитом, где второй общий адаптер включает второй сайт связывания с праймером. Обработанные бисульфитом фрагменты нуклеиновой кислоты-мишени, иммобилизованные на твердом носителе, подвергают амплификации и получают секвенирующую библиотеку для определения статуса метилирования нуклеиновой кислоты-мишени.
В одном из аспектов изобретения описаны способы получения секвенирующей библиотеки для определения статуса метилирования нуклеиновой кислоты-мишени. Эти способы включают получение множества твердых носителей, содержащих транспозомные комплексы, иммобилизованные на этих носителях. Транспозомные комплексы включают транспозоны и транспозазы, где транспозоны содержат перенесенные цепи и неперенесенные цепи. Перенесенная цепь включает (i) первую часть, расположенную у 3'-конца и содержащую последовательность распознавания транспозазы, и (ii) вторую часть, расположенную у 5'-конца по отношению к первой части, содержащей первую последовательность адаптера и первый член связывающейся пары. Первый член связывающейся пары связывается со вторым членом связывающейся пары на твердом носителе, что приводит к иммобилизации транспозона на твердом носителе. Первый адаптер также включает первую последовательность, связывающуюся с праймером. Неперенесенная цепь включает (i) первую часть, расположенную у 5'-конца и содержащую последовательность распознавания транспозазы, и (ii) вторую часть, расположенную у 3'-конца по отношению к первой части, содержащей вторую последовательность адаптера, где концевой нуклеотид у 3'-конца является блокированным. Второй адаптер также включает вторую последовательность, связывающуюся с праймером. Нуклеиновую кислоту-мишень подвергают контактированию со множеством твердых носителей, содержащих иммобилизованные транспозомные комплексы. Нуклеиновую кислоту-мишень фрагментируют с образованием множества фрагментов, и множество перенесенных цепей встраивают в 5'-конец по меньшей мере одной цепи фрагментов, что приводит к иммобилизации фрагментов нуклеиновой кислоты-мишени на твердом носителе. 3'-конец фрагментированной нуклеиновой кислоты-мишени удлиняют с использованием ДНК-полимеразы. Неперенесенную цепь лигируют с 3'-концом фрагментированной нуклеиновой кислоты-мишени. Затем иммобилизованные фрагменты нуклеиновой кислоты-мишени подвергают обработке бисульфитом. 3'-конец иммобилизованных фрагментов нуклеиновой кислоты-мишени, разрушенных в процессе обработки бисульфитом, удлиняют с использованием ДНК-полимеразы так, чтобы 3'-конец иммобилизованных фрагментов нуклеиновой кислоты-мишени содержал гомополимерный «хвост». Вторую последовательность адаптера вводят в 3'-конец иммобилизованных фрагментов нуклеиновой кислоты-мишени, разрушенных в процессе обработки бисульфитом. Обработанные бисульфитом фрагменты нуклеиновой кислоты-мишени, иммобилизованные на твердом носителе, подвергают амплификации с использованием первого и второго праймера, в результате чего получают секвенирующую библиотеку для определения статуса метилирования нуклеиновой кислоты-мишени.
В одном из аспектов изобретения описаны способы получения секвенирующей библиотеки для определения статуса метилирования нуклеиновой кислоты-мишени. Эти способы включают контактирование нуклеиновой кислоты-мишени с транспозомными комплексами, содержащими транспозоны и транспозазы. Транспозоны содержат перенесенные цепи и неперенесенные цепи. Перенесенная цепь включает (i) первую часть, расположенную у 3'-конца и содержащую последовательность распознавания транспозазы, и (ii) вторую часть, расположенную у 5'-конца по отношению к первой части, содержащей первую последовательность адаптера и первый член связывающейся пары, где первый член связывающейся пары связывается со вторым членом связывающейся пары. Неперенесенная цепь включает (i) первую часть, расположенную у 5'-конца и содержащую последовательность распознавания транспозазы, и (ii) вторую часть, расположенную у 3'-конца по отношению к первой части, содержащей вторую последовательность адаптера, где концевой нуклеотид у 3'-конца является блокированным, и где второй адаптер включает вторую последовательность, связывающуюся с праймером. Нуклеиновую кислоту-мишень фрагментируют с образованием множества фрагментов, и множество перенесенных цепей встраивают в 5'-конец по меньшей мере одной цепи фрагментов, что приводит к иммобилизации фрагментов нуклеиновой кислоты-мишени на твердом носителе. Фрагменты нуклеиновой кислоты-мишени, содержащие транспозонный конец, подвергают контактированию со множеством твердых носителей, содержащих второй член связывающейся пары, где связывание первого члена связывающейся пары со вторым членом связывающейся пары приводит к иммобилизации нуклеиновой кислоты-мишени на твердом носителе. 3'-конец фрагментированной нуклеиновой кислоты-мишени удлиняют с использованием ДНК-полимеразы. Неперенесенную цепь лигируют с 3'-концом фрагментированной нуклеиновой кислоты-мишени. Иммобилизованные фрагменты нуклеиновой кислоты-мишени подвергают обработке бисульфитом. 3'-конец иммобилизованных фрагментов нуклеиновой кислоты-мишени, разрушенных в процессе обработки бисульфитом, удлиняют с использованием ДНК-полимеразы так, чтобы 3'-конец иммобилизованных фрагментов нуклеиновой кислоты-мишени содержал гомополимерный «хвост». Вторую последовательность адаптера вводят в 3'-конец иммобилизованных фрагментов нуклеиновой кислоты-мишени, разрушенных в процессе обработки бисульфитом. Обработанные бисульфитом фрагменты нуклеиновой кислоты-мишени, иммобилизованные на твердом носителе, подвергают амплификации с использованием первого и второго праймера, в результате чего получают секвенирующую библиотеку для определения статуса метилирования нуклеиновой кислоты-мишени.
В некоторых вариантах осуществления изобретения, концевой нуклеотид у 3'-конца второго адаптера блокируют членом, выбранным из группы, состоящей из дидезокси-нуклеотида, фосфатной группы, тиофосфатной группы и азидогруппы.
В некоторых вариантах осуществления изобретения, аффинные молекулы могут быть членами связывающейся пары. В некоторых случаях, модифицированные нуклеиновые кислоты могут содержать первый член связывающейся пары, а зонд для захвата может содержать второй член связывающейся пары. В некоторых случаях, зонды для захвата могут быть иммобилизованы на твердой поверхности, где модифицированная нуклеиновая кислота может содержать первый член связывающейся пары, а зонд для захвата может содержать второй член связывающейся пары. В таких случаях, связывание первого и второго членов связывающейся пары приводит к иммобилизации модифицированной нуклеиновой кислоты на твердой поверхности. Примерами связывающихся пар являются, но не ограничиваются ими, биотин-авидин, биотин-стрептавидин, биотин-нейтравидин, лиганд-рецептор, гормон-рецептор, лектин-гликопротеин, олигонуклеотид-комплементарный олигонуклеотид и антиген-антитело.
В некоторых вариантах осуществления изобретения, первую общую последовательность адаптера встраивают в 5'-конец фрагментов нуклеиновой кислоты-мишени посредством односторонней транспозиции. В некоторых вариантах осуществления изобретения, первую общую последовательность адаптера встраивают в 5'-конец фрагментов нуклеиновой кислоты-мишени посредством лигирования. В некоторых вариантах осуществления изобретения, введение второй общей последовательности адаптера в обработанные бисульфитом иммобилизованные фрагменты нуклеиновой кислоты-мишени включает (i) удлинение 3'-конца иммобилизованных фрагментов нуклеиновой кислоты-мишени с использованием концевой трансферазы так, чтобы они содержали гомополимерный «хвост»; (ii) гибридизацию олигонуклеотида, содержащего одноцепочечную гомополимерную часть и двухцепочечную часть, включающую вторую общую последовательность адаптера, где одноцепочечная гомополимерная часть комплементарна гомополимерному «хвосту»; и (iii) лигирование второй общей последовательности адаптера с иммобилизованными фрагментами нуклеиновой кислоты-мишени для включения второй общей последовательности адаптера в обработанные бисульфитом иммобилизованные фрагменты нуклеиновой кислоты-мишени.
В некоторых вариантах осуществления изобретения, нуклеиновую кислоту-мишень выделяют из одной клетки. В некоторых вариантах осуществления изобретения, нуклеиновую кислоту-мишень выделяют из одной органеллы. В некоторых вариантах осуществления изобретения, нуклеиновой кислотой-мишенью является геномная ДНК. В некоторых вариантах осуществления изобретения, нуклеиновую кислоту-мишень подвергают перекрестному связыванию с другими нуклеиновыми кислотами. В некоторых вариантах осуществления изобретения, нуклеиновую кислоту-мишень выделяют из залитого в парафин образца, фиксированного формалином (FFPE). В некоторых вариантах осуществления изобретения, нуклеиновую кислоту-мишень подвергают перекрестному связыванию с белками. В некоторых вариантах осуществления изобретения, нуклеиновую кислоту-мишень подвергают перекрестному связыванию с ДНК. В некоторых вариантах осуществления изобретения, нуклеиновой кислотой-мишенью является ДНК, защищенная гистоном. В некоторых вариантах осуществления изобретения, гистоны удаляют из нуклеиновой кислоты-мишени. В некоторых вариантах осуществления изобретения, нуклеиновой кислотой-мишенью является неклеточная опухолевая ДНК. В некоторых вариантах осуществления изобретения, неклеточную опухолевую ДНК выделяют из плацентарной жидкости. В некоторых вариантах осуществления изобретения, неклеточную опухолевую ДНК выделяют из плазмы. В некоторых вариантах осуществления изобретения, плазму выделяют из цельной крови с использованием мембранного сепаратора, имеющего зону сбора плазмы. В некоторых вариантах осуществления изобретения, зона для сбора плазмы включает транспозомные комплексы, иммобилизованные на твердом носителе. В некоторых вариантах осуществления изобретения, нуклеиновой кислотой-мишенью является кДНК. В некоторых вариантах осуществления изобретения, твердым носителем является сфера. В некоторых вариантах осуществления изобретения, множество твердых носителей представляет собой множество сфер, имеющих различные размеры.
В некоторых вариантах осуществления изобретения, одна последовательность со штрих-кодом присутствует во множестве олигонуклеотидов, иммобилизованных на каждом отдельном твердом носителе. В некоторых вариантах осуществления изобретения, различные последовательности со штрих-кодом присутствуют во множестве олигонуклеотидов, иммобилизованных на каждом отдельном твердом носителе. В некоторых вариантах осуществления изобретения, перенос информации о последовательности со штрих-кодом во фрагменты нуклеиновой кислоты-мишени осуществляют путем лигирования. В некоторых вариантах осуществления изобретения, перенос информации о последовательности со штрих-кодом во фрагменты нуклеиновой кислоты-мишени осуществляют путем удлинения с использованием полимеразы. В некоторых вариантах осуществления изобретения, перенос информации о последовательности со штрих-кодом во фрагменты нуклеиновой кислоты-мишени осуществляют путем лигирования и удлинения с использованием полимеразы. В некоторых вариантах осуществления изобретения, удлинение с использованием полимеразы осуществляют посредством удлинения 3'-конца нелигированной транспозонной цепи посредством ДНК-полимеразы с использованием лигированного иммобилизованного олигонуклеотида в качестве матрицы. В некоторых вариантах осуществления изобретения, по меньшей мере часть последовательностей адаптера также содержит вторую последовательность со штрих-кодом.
В некоторых вариантах осуществления изобретения, транспозомные комплексы являются мультимерными, где последовательности адаптера транспозонов каждой мономерной единицы отличаются от последовательностей другой мономерной единицы, присутствующей в том же самом транспозомном комплексе. В некоторых вариантах осуществления изобретения, последовательность адаптера также включает первую последовательность, связывающуюся с праймером. В некоторых вариантах осуществления изобретения, первый сайт связывания с праймером имеет последовательность, которая не является гомологичной последовательности для захвата или ее комплементу. В некоторых вариантах осуществления изобретения, олигонуклеотиды, иммобилизованные на твердом носителе, также содержат вторую последовательность, связывающуюся с праймером.
В некоторых вариантах осуществления изобретения, транспозомные комплексы являются мультимерными, где транспозонные мономерные единицы связаны друг с другом в одном и том же транспозомном комплексе. В некоторых вариантах осуществления изобретения, транспозаза транспозомной мономерной единицы связана с транспозазой другой транспозомной мономерной единицы одного и того же транспозомного комплекса. В некоторых вариантах осуществления изобретения, транспозоны транспозомной мономерной единицы связаны с транспозонами другой транспозомной мономерной единицы одного и того же транспозомного комплекса. В некоторых вариантах осуществления изобретения, транспозаза транспозомной мономерной единицы связана с транспозазой другой транспозомной мономерной единицы одного и того же транспозомного комплекса посредством ковалентной связи. В некоторых вариантах осуществления изобретения, транспозазы одной мономерной единицы связаны с транспозазой другой транспозомной мономерной единицы одного и того же транспозомного комплекса посредством дисульфидной связи. В некоторых вариантах осуществления изобретения, транспозоны транспозомной мономерной единицы связаны с транспозонами другой транспозомной мономерной единицы одного и того же транспозомного комплекса посредством ковалентной связи.
В некоторых вариантах осуществления изобретения, информация о сцеплении последовательности нуклеиновой кислоты-мишени представляет собой информацию о гаплотипе. В некоторых вариантах осуществления изобретения, информация о сцеплении последовательности нуклеиновой кислоты-мишени представляет информацию о геномных вариантах. В некоторых вариантах осуществления изобретения, геномные варианты выбраны из группы, состоящей из делеций, транслокаций, межхромосомных сцеплений генов, дупликаций и паралогов. В некоторых вариантах осуществления изобретения, олигонуклеотиды, иммобилизованные на твердом носителе, содержат частично двухцепочечную область и частично одноцепочечную область. В некоторых вариантах осуществления изобретения, частично одноцепочечная область олигонуклеотида содержит вторую последовательность со штрих-кодом и вторую последовательность, связывающуюся с праймером. В некоторых вариантах осуществления изобретения, фрагменты нуклеиновой кислоты-мишени, имеющие штрих-коды, амплифицируют, а затем определяют последовательность фрагментов нуклеиновой кислоты-мишени. В некоторых вариантах осуществления изобретения, последующую амплификацию проводят в одном реакционном сосуде до определения последовательности фрагментов нуклеиновой кислоты-мишени. В некоторых вариантах осуществления изобретения, третью последовательность со штрих-кодом вводят во фрагменты нуклеиновой кислоты-мишени во время амплификации.
В некоторых вариантах осуществления изобретения, способы могут также включать объединение фрагментов нуклеиновой кислоты-мишени, содержащих штрих-коды из первой серии множества реакционных сосудов, в пул фрагментов нуклеиновой кислоты-мишени, содержащих штрих-коды; перераспределение пула фрагментов нуклеиновой кислоты-мишени, содержащих штрих-коды, с получением множества второй серии множества реакционных сосудов; и введение третьего штрих-кода во фрагменты нуклеиновой кислоты-мишени путем амплификации фрагментов нуклеиновой кислоты-мишени во вторую серию множества реакционных сосудов до секвенирования.
В некоторых вариантах осуществления изобретения, способы могут также включать предварительное фрагментирование нуклеиновой кислоты-мишени до контактирования нуклеиновой кислоты-мишени с транспозомными комплексами. В некоторых вариантах осуществления изобретения, предварительное фрагментирование нуклеиновой кислоты-мишени представляет собой способ, выбранный из группы, состоящей из обработки ультразвуком и расщепления рестриктирующими ферментами.
Краткое описание чертежей
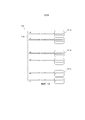
На фигуре 1 представлена блок-схема, на которой проиллюстрирован метод связывания транспозом с поверхностью сфер.
На фигуре 2 проиллюстрированы стадии способа, описанного на фигуре 1.
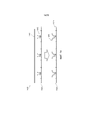
На фигуре 3 схематически представлена диаграмма, иллюстрирующая способ тагментации на поверхности сфер.
На фигуре 4 представлена таблица данных о выходах ДНК для различных кластров в способе тагментации на сферах, как показано на фигуре 3.
На фигуре 5 представлена таблица данных, полученных для другого примера воспроизводимости способа тагментации на сферах одинакового размера как показано на фигуре 3.
На фигурах 6A и 6B представлен график размера вставок для пула 1 и график размера вставок для пула 2 индексированных образцов на фигуре 5, соответственно.
На фигуре 7 представлена гистограмма воспроизводимости общего числа ридов и процента ридов, выровненных для проведения эксперимента, как показано на фигуре 5.
На фигурах 8A, 8B и 8C представлен график данных о размерах вставки в контрольной библиотеке, график данных о размерах вставки в тагментированной библиотеке на сферах, а также представлена таблица, в которой систематизированы данные, полученные в анализе на обогащение экзома, соответственно.
На фигурах 9A, 9B и 9C представлены гистограмма дупликаций PF, гистограмма для фракции отобранных оснований и гистограмма для оснований, используемых в PCT на мишени, соответственно, в анализе на обогащение экзома.
На фигуре 10 представлена блок-схема, иллюстрирующая репрезентативный метод продуцирования транспозомных комплексов на поверхности сфер.
На фигурах 11, 12 и 13 проиллюстрированы стадии метода, описанного на фигуре 10.
На фигуре 14 схематически представлена диаграмма способа тагментации, проводимого с использованием сферы, покрытой транспозомой как показано на фигуре 13.
На фигуре 15 представлена репрезентативная схема образования транспозом на твердом носителе.
На фигуре 16 представлена репрезентативная схема получения библиотек сцепленных нуклеиновых кислот с уникальными индексами.
На фигуре 17 представлена репрезентативная схема получения библиотек сцепленных нуклеиновых кислот с уникальными индексами.
На фигурах 18 и 19 проиллюстрированы захват одной CPT-ДНК на одной клональной индексированной сфере, где CPT-ДНК обертывает эту сферу.
На фигуре 20 схематически проиллюстрировано связывание Y-адаптера, иммобилизованного на твердой поверхности, с тагментированной ДНК посредством лигирования и заполнения брешей.
На фигуре 21 схематически проиллюстрировано получение таких Y-адаптеров в процессе лигирования CPT-DNA с олигонуклеотидами, иммобилизованными на твердом носителе.
На фигуре 22 проиллюстрирован электрофорез в агарозном геле для удаления свободной транспозомы из библиотек сцепленных нуклеиновых кислот с помощью эксклюзионной хроматографии.
На фигуре 23 представлена репрезентативная схема получения библиотеки последовательностей специфических фрагментов ДНК методом «дробовика».
На фигуре 24 представлена репрезентативная схема сбора информации о последовательности клональной индексироыванной секвенирующей библиотеки.
На фигуре 25 представлены результаты оптимизации плотности зондов для захвата на сферах.
На фигуре 26 представлены результаты теста на возможность получения индексированных секвенирующих библиотек CPT-ДНК на сферах путем внутримолекулярной гибридизации.
На фигуре 27 представлены результаты теста на возможность клонального индексирования.
На фигуре 28 представлен график, на котором проиллютрирована частота секвенирующих ридов для конкретных расстояний в островках (внутри островков), а также между соседними выровненными островками ридов для матричной нуклеиновой кислоты после тагментации.
На фигурах 29A и 29B представлены репрезентативные методы получения информации о сцеплении генов на твердом носителе.
На фигурах 30 и 31 схематически представлены транспозиция индексированных клонированных сфер в одном реакционном сосуде (в одном сосуде) и результаты транспозиции.
На фигуре 32 схематически проиллюстрировано создание клональных транспозом на сферах с использованием 5'- или 3'-биотинилированных олигонуклеотидов.
На фигуре 33 показаны размеры библиотеки для транспозом на сферах.
На фигуре 34 показано влияние поверхностной плотности транспозом на размер вставки.
На фигуре 35 показано влияние исходной ДНК на размер распределения.
На фигуре 36 показаны размеры островков и распределение, достигаемое посредством реакции тагментации на сферах и в растворе.
На фигуре 37 проиллюстрировано клональное индексирование нескольких отдельных молекул ДНК, каждая из которых имеет уникальные индексы.
На фигуре 38 представлена диаграмма для устройства, применяемого в целях выделения плазмы из цельной крови.
На фигурах 39 и 40 представлена диаграмма для устройства, применяемого в целях выделения плазмы и последующего использования выделенной плазмы.
На фигуре 41 представлена репрезентативная схема целевого фазирования посредством обогащения специфических областей генома.
На фигуре 42 представлена репрезентативная схема фазирования экзома с использованием SNP, расположенных между экзонами.
На фигуре 43 представлена репрезентативная схема одновременного фазирования и детектирования метилирования.
На фигуре 44 представлена альтернативная репрезентативная схема одновременного фазирования и детектирования метилирования.
На фигуре 45 представлена репрезентативная схема получения библиотек различных размеров с использованием клонально индексированных сфер различных размеров в одном анализе.
На фиг. 46 представлена репрезентативная схема определения генетических вариантов библиотек, имеющих длины различных масштабов.
На фиг. 47 A и B представлены резульаты детектирования гомозиготной 60 т.п.о.-делеции в хромосоме 1.
На фиг. 48 представлены резульаты детектирования сцепления генов способами согласно изобретению.
На фиг. 49 представлены результаты детектирования генетических делеций способами согласно изобретению.
На фиг. 50 представлены ME-последовательности до и после превращения под действием бисульфита.
На фиг. 51 представлены результаты оптимизации эффективности превращения под действием бисульфита.
На фиг. 52 представлены результаты превращения под действием бисульфита на графике IVC (на графике зависимости интенсивности от циклов на одно основание).
На фиг. 53 представлены изображения, полученные с помощью электрофореза в агарозном геле для сцепленных с индексами библиотек после проведения ПЦР, осуществляемой после превращения под действием бисульфита (BSC).
На фиг. 54 проиллюстрировано мечение сцепленных с индексами библиотек CPT-seq с помощью биоанализатора после обогащения этих библиотек без отбора по размеру.
На фиг. 55 проиллюстрирован анализ библиотек в агарозном геле после обогащения.
На фиг. 56 представлены результаты применения целевого гаплотипирования в области HLA в хромосоме.
На фиг. 57 проиллюстрированы некоторые возможные механизмы замены ME.
На фиг. 58 проиллюстрированы некоторые возможные механизмы замены ME.
На фиг. 59 представлена часть транспозазы Tn5, в которой репрезентативные аминокислотные остатки Asp468, Tyr407, Asp461, Lys459, Ser458, Gly462, Ala466, Met470 могут быть заменены Cys.
На фиг. 60 представлена часть транспозазы Tn5 с аминокислотными заменами S458C, K459C и A466C, введенными так, чтобы цистеиновые остатки могли образовывать дисульфидную связь между двумя мономерными единицами.
На фиг. 61 представлена репрезентативная схема получения и использования биоконъюгата «димерная транспозаза (dTnp)-наночастица (NP)» (dTnp-NP) посредством наночастиц, покрытых амином.
На фиг. 62 представлена репрезентативная схема конъюгирования транспозомного димера с твердым носителем, покрытым амином.
На фиг. 63 представлен транспозомный комплекс Mu, в котором транспозонные концы являются сцепленными.
На фиг. 64 представлена диаграмма индексированных сцепленных ридов для сборки/фазирования псевдогенов и показано преимущество идентификации вариантов в псевдогене с использованием коротких фрагментов.
На фиг. 65 проиллюстрирован график замен индексов для 4 отдельных экспериментов, где такая замена представлена как % замененных индексов.
На фиг. 66 проиллюстрирован анализ размеров фрагментов, проведенный посредством титрования Ts-Tn5 на биоанализаторе Agilent BioAnalyzer.
На фиг. 67 представлена репрезентативная схема повышения выхода ДНК в соответствии с протоколом Epi-CPTSeq с применением ферментативных методов восстановления разрушенных элементов библиотеки после обработки бисульфитом.
На фиг. 68 A-C представлено несколько репрезентативных схем повышения выхода ДНК в соответствии с протоколом Epi-CPTSeq с применением ферментативных методов восстановления разрушенных элементов библиотеки после обработки бисульфитом.
На фиг. 69 представлена репрезентативная схема «спасения» матрицы методом рандомизированного удлиненеия праймера.
На фиг. 70 проиллюстрирована фрагментация библиотеки ДНК в процессе реакции превращения с использованием бисульфата натрия. На левой панели проиллюстрирована фрагментация в процессе превращения части ДНК, тагментированной на магнитных сферах, посредством бисульфата. На правой панели показаны следовые количества библиотек CPTSeq и Epi-CPTSeq (Me-CPTSeq), оцененные с помощью биоанализатора BioAnalyzer.
На фиг. 71 представлена репрезентативная схема и результаты TdT-опосредуемой реакции лигирования оцДНК.
На фиг. 72 представлены схемы и результаты TdT-опосредуемого восстановления связанной со сферой библиотеки после превращения посредством бисульфата натрия. На левой панели проиллюстрирована технологическая схема «спасения» библиотеки ДНК посредством бисульфитного превращения с использованием TdT-опосредуемой реакции лигирования. Результаты эксперимента по «спасению» библиотеки ДНК представлены на правой панели.
На фиг. 73 представлены результаты анализа на метилирование СPTSeq.
На фиг. 74 представлена репрезентативная схема превращения ДНК, присутствующей на сферах, посредством бисульфита.
На фиг. 75 A-B представлены результаты оптимизации эффективности после превращения посредством бисульфита.
Подробное описание изобретения
Варианты настоящего изобретения относятся к секвенированию нуклеиновых кислот. В частности, описанные здесь варианты способов и композиций относятся к получению матриц на основе нуклеиновых кислот и к получению данных для этих последовательностей.
В одном из своих аспектов, настоящее изобретение относится к способам тагментации (фрагментирования и мечения) нуклеиновой кислоты-мишени на твердом носителе для конструирования тагментированной библиотеки нуклеиновой кислоты-мишени. В одном из вариантов осуществления изобретения, твердым носителем являются сферы. В одном из вариантов осуществления изобретения, нуклеиновой кислоты-мишенью является ДНК.
В одном из своих аспектов, настоящее изобретение относится к способам и композициям твердого носителя, а также к разработанным на основе транспозазы способам, которые позволяют получить информацию о сцеплении нуклеиновой кислоты-мишени. В некоторых вариантах осуществления изобретения, указанные композиции и способы позволяют получить информацию о сборке/фазировании.
В одном из своих аспектов, настоящее изобретение относится к способам и композициям, применяемым для получения информации о сцеплении генов посредством захвата сцепленных и перенесенных нуклеиновых кислот-мишеней на твердом носителе.
В одном из аспектов изобретения, описанные здесь композиции и способы применяются для анализа геномных вариантов. Репрезентативными геномными вариантами являются, но не ограничиваются ими, делеции, межхромосомные транслокации, дупликации, паралоги, межхромосомные сцепления генов. В некоторых вариантах осуществления изобретения, описанные здесь композиции и способы применяются для получения информации о фазировании геномных вариантов.
В одном из аспектов изобретения, описанные здесь композиции и способы применяются для фазирования специфических областей нуклеиновой кислоты-мишени. В одном из вариантов осуществления изобретения, нуклеиновой кислотой-мишенью является ДНК. В одном из вариантов осуществления изобретения, нуклеиновой кислотой-мишенью является геномная ДНК. В некоторых вариантах осуществления изобретения, нуклеиновой кислотой-мишенью является РНК. В некоторых вариантах осуществления изобретения, РНК представляет собой мРНК. В некоторых вариантах осуществления изобретения, нуклеиновой кислотой-мишенью является комплементарная ДНК (кДНК). В некоторых вариантах осуществления изобретения, нуклеиновая кислота-мишени выделена из одной клетки. В некоторых вариантах осуществления изобретения, нуклеиновая кислота-мишени выделена из опухолевых клеток кровотока. В некоторых вариантах осуществления изобретения, нуклеиновой кислотой-мишенью является неклеточная ДНК. В некоторых вариантах осуществления изобретения, нуклеиновой кислотой-мишенью является неклеточная опухолевая ДНК. В некоторых вариантах осуществления изобретения, нуклеиновая кислота-мишень выделена из залитых из залитых в парафин образцов ткани, фиксированных формалином. В некоторых вариантах осуществления изобретения, нуклеиновой кислотой-мишенью является перекрестно связанная нуклеиновая кислота-мишень. В некоторых вариантах осуществления изобретения, нуклеиновая кислота-мишень перекрестно связана с белками. В некоторых вариантах осуществления изобретения, нуклеиновая кислота-мишень перекрестно связана с нуклеиновой кислотой. В некоторых вариантах осуществления изобретения, нуклеиновой кислотой-мишенью является ДНК, защищенная гистоном. В некоторых вариантах осуществления изобретения, защищенную гистоном ДНК осаждают из клеточного лизата с использованием антител против гистонов, а затем гистоны удаляют.
В некоторых аспектах изобретения, индексированные библиотеки создают из нуклеиновой кислоты-мишени с использованием клонально индексированных сфер. В некоторых вариантах осуществления изобретения, тагментированная нуклеиновая кислота-мишень, в случае когда ДНК-мишень еще связана с транспозазой, может быть захвачена с использованием клонально индексированных сфер. В некоторых вариантах осуществления изобретения, специфические зонды для захвата используют в целях захвата специфической представляющей интерес области в нуклеиновой кислоте-мишени. Захваченные области нуклеиновой кислотой-мишени могут быть промыты в различных условиях жесткости, а затем амплифицированы, но необязательно, и секвенированы. В некоторых вариантах осуществления изобретения, зонд для захвата может быть биотинилированным. Комплекс биотинилированных зондов для захвата, гибридизованных со специфическими областями индексированных нуклеиновых кислот-мишеней, может быть разделен с использованием стрептавидиновых сфер. Репрезентативная схема целевого фазирования представлена на фиг. 41.
В некоторых аспектах изобретения, описанные здесь композиции и способы могут быть применены для фазирования экзонов. В некоторых вариантах осуществления изобретения, экзоны и промоторы могут быть обогащены. Маркеры, например, гетерозиготные SNP, находящиеся между экзонными областями, могут облегчать фазирование экзонов, а в частности, если между экзонами имеется большое расстояние. Репрезентативное фазирование экзонов проиллюстрировано на фиг. 42. В некоторых вариантах осуществления изобретения, индексированные сцепленные риды не могут одновременно охватывать (покрывать) гетерозиготные SNP соседних экзонов. Таким образом, это затрудняет фазирование двух или более экзонов. Описанные здесь композиции и способы также применяются для обогащения гетерозиготных SNP, расположенных между экзованми, например, для фазирования экзона 1 на SNP1, и SNP2 на экзон 2. Таким образом, с использованием SNP 1 может быть проведено фазирование экзона 1 и экзона 2, как показано на фиг. 42.
В одном из аспектов изобретения, описанные здесь композиции и способы могут быть применены для одновременного фазирования и детектирования метилирования. Детектирование метилирования посредством превращения в результате обработки бисульфитом (BSC) является проблематичным, поскольку реакция BSC является слишком жесткой для ДНК, то есть, приводит к фрагментации ДНК, а поэтому она удаляет информацию о сцеплении/фазировании. Способы, описанные в настоящей заявке, имеют дополнительное преимущество, заключающееся в том, что при их проведении не требуется дополнительной стадии очистки как в традиционных методах BSC, и это способствует увеличению выхода.
В одном из аспектов изобретения, описанные здесь композиции и способы могут быть применены для получения библиотек различных размеров в одном анализе. В некоторых вариантах осуществления изобретения, клонально индексированные сферы различных размеров могут быть использованы для получения библиотек различных размеров. На фигуре 1 проиллюстрирована блок-схема метода 100, применяемого для связывания транспозом с поверхностью сфер. Транспозомы могут быть присоединены к поверхности сфер любым химическим методом, который может быть осуществлен на олигонуклеотиде транспозона, транспозазе и твердой фазе. В одном из примеров, транспозомы связаны с поверхностью сфер посредством комплекса «биотин-стрептавидин». Метод 100 включает, но не ограничивается ими, нижеследующие стадии.
В одном из вариантов осуществления изобретения, транспозоны могут содержать секвенирующие сайты связывания с праймером. Репрезентативными последовательностями сайтов связывания с последовательностью являются, но не ограничиваются ими, AATGATACGGCGACCACCGAGATCTACAC (последовательность P5) и CAAGCAGAAGACGGCATACGAGAT (последовательность P7). В некоторых вариантах осуществления изобретения, транспозоны могут быть биотинилированными.
В стадии 110, показанной на фигуре 1, были получены биотинилированные транспозоны P5 и P7. Эти транспозоны могут также включать одну или более индексных последовательностей (уникальный идентификатор). Репрезентативными индексными последовательностями являются, но не ограничиваются ими, TAGATCGC, CTCTCTAT, TATCCTCT, AGAGTAGA, GTAAGGAG, ACTGCATA, AAGGAGTA, CTAAGCCT. В другом примере, биотинилированными являются только транспозоны P5 или только транспозоны P7. В другом примере, транспозоны включают только мозаичные концевые (ME) последовательности или последовательности ME плюс дополнительные последовательности, которые не являются последовательностями P5 и P7. В этом примере, последовательности P5 и P7 добавляют в последующей стадии ПЦР-амплификации.
В стадии 115, показанной на фигуре 1, была осуществлена сборка транспозом. Собранные транспозомы представляют собой смесь транспозом P5 и P7. Смесь транспозом P5 и P7 более подробно описаны со ссылкой на фигуры 11 и 12.
В стадии 120, показанной на фигуре 1, смеси транспозом P5/P7 связаны с поверхностью сфер. В этом примере, сферы представляют собой сферы, покрытые стрептавидином, а транспозомы связаны с последовательностью сфер посредством комплекса «биотин-стрептавидин». Сферы имеют различные размеры. В одном из примеров, размер сфер может составлять 2,8 мкм. В другом примере, размер сфер может составлять 1 мкм. Суспензия (например, 1 мкл) сфер размером 1 мкм имеет большую площадь поверхности на объем связывающихся транспозом. Благодаря доступности площади поверхности для связывания транспозом, число продуктов тагментирования на реакцию увеличивается.
На фигуре 2 проиллюстрированы стадии 110, 115 и 120 метода, показанного на фигуре 1. В этом примере, транспозоны представлены в виде дуплексов. В другом примере (не показаны) может быть использована другая структура, такая как шпилька, то есть, один олигонуклеотид с аутокомплементарными областями, способными образовывать деплекс.
В стадии 110 метода 100 было получено множество биотинилированных транспозонов P5 210a и множество транспозонов P7 210b. Транспозоны P5 210a и транспозоны P7 210b были биотинилированными.
В стадии 115 метода 100, транспозоны P5 210a и транспозоны P7 210b смешивают с транспозазой Tn5 215 с получением множества собранных транспозом 220.
В стадии 120 метода 100, транспозомы 220 связаны со сферой 225. Сферой 225 является сфера, покрытая стрептавидином. Транспозомы 220 связаны со сферой 225 посредством комплекса «биотин-стрептавидин».
В одном из вариантов осуществления изобретения, смесь транспозом может быть получена на твердом носителе, таком как поверхность сферы как показано на фигурах 10, 11, 12 и 13. В этом примере, олигонуклеотиды P5 и P7 сначала связывают с поверхностью сферы, а затем осуществляют сборку транспозомных комплексов.
На фигуре 3 схематически проиллюстрирована диаграмма репрезентативного способа тагментации 300 на поверхности сферы. На этой фигуре проиллюстрирован способ 300 для сферы 225 со связанными с ней транспозомами 220, как показано на фигуре 2. Раствор ДНК 310 добавляют к суспензии сфер 225. Поскольку ДНК 310 контактирует с транспозомами 220, то ДНК является тагментированной (фрагментированной и меченной) и связанной со сферами 225 посредством транспозом 220. Связанная и тагментированная ДНК 310 может быть подвергнута ПЦР-амплификации с получением пула ампликонов 315 в растворе (без сфер). Ампликоны 315 могут быть перенесены на поверхность проточной кюветы 320. Протокол получения кластера (например, протокол мостиковой амплификации или протокол любой другой амплификации, который может быть проведен для создания кластера) может быть проведен для получения множества кластеров 325 на поверхности проточной кюветы 320. Кластеры 325 представляют собой продукты клональной амплификации тагментированнй ДНК 310. Кластеры 325 уже готовы для проведения следующей стадии протокола секвенирования.
В другом варианте осуществления изобретения, транспозомы могут быть связаны с любой твердой поверхностью, такой как стенки микроцентрифужной пробирки.
В другом варианте получения смеси транспозомных комплексов на поверхности сфер, олигонуклеотиды сначала связывают с поверхностью сферы, а затем осуществляют сборку транспозомы. На фигуре 10 проиллюстрирована блок-схема репрезентативного метода 1000, применяемого для создания транспозомных комплексов на поверхности сферы. Метод 1000 включает, но не ограничивается ими, нижеследующие стадии.
В стадии 1010, олигонуклеотиды P5 и P7 связывают с поверхностью сферы. В одном из примеров, олигонуклеотиды P5 и P7 являются биотинилированными, а сферой является сфера, покрытая стрептавидином. Эта стадия также схематически проиллюстрирована на диаграмме 1100 фигуры 11. Как показано на фигуре 11, олигонуклеотид P5 1110 и олигонуклеотид P7 1115 связаны с поверхностью сферы 1120. В этом примере, один олигонуклеотид P5 1110 и один олигонуклеотид P7 1115 связаны с поверхностью сферы 1120, однако, с поверхностью множества сфер 1120 могут быть связаны олигонуклеотиды P5 1110 и/или олигонуклеотиды P7 1115 в любом количестве. В одном из примеров, олигонуклеотид P5 1110 содержит последовательность праймера P5, индексную последовательность (уникальный идентификатор), секвенирующую последовательность праймера рида 1 и мозаичную концевую последовательность (ME). В этом примере, олигонуклеотид P7 1115 содержит последовательность праймера P7, индексную последовательность (уникальный идентификатор), секвенирующую последовательность праймера рида 2 и последовательность ME. В другом примере (не показано), индексная последовательность присутствует только в олигонуклеотиде P5 1110. В другом примере (не показано), индексная последовательность присутствует только в олигонуклеотиде P7 1115. В еще одном примере (не показано), индексная последовательность отсутствует в олигонуклеотиде P5 1110 и в олигонуклеотиде P7 1115.
В стадии 1015, комплементарные мозаичные концевые (ME′) олигонуклеотиды гибридизуют с олигонуклеотидами P5 и P7, связанными со сферами. Эта стадия также схематически проиллюстрирована на диаграмме 1200 на фигуре 12. Как показано на фигуре 12, комплементарные ME-последовательности (ME′) 1125 гибридизованы с олигонуклеотидом P5 1110 и с олигонуклеотидом P7 1115. Комплементарные ME-последовательности (ME′) 1125 (например, комплементарные ME-последовательности (ME′) 1125a и комплементарные ME-последовательности (ME′) 1125b) гибридизованы с ME-последовательностями олигонуклеотида P5 1110 и олигонуклеотида P7 1115, соответственно. Комплементарные ME-последовательности (ME′) 1125 обычно имеют длину приблизительно 15 оснований и фосфорилированы у 5'-конца.
В стадии 1020, фермент транспозазу добавляют к олигонуклеотидам, связанным со сферами, с образованием смеси связанных со сферами транспозомных комплексов. Эта стадия также схематически проиллюстрирована на диаграмме 1300 на фигуре 13. Как показано на фигуре 13, фермент транспозазу добавляют с образованием множества транспозомных комплексов 1310. В этом примере, транспозомным комплексом 1310 является дуплексная структура, содержащая фермент транспозазу и две связанных с поверхностью олигонуклеотидных последовательности, а также гибридизованные с ними комплементарные ME-последовательности (ME′) 1125. Так, например, транспозомный комплекс 1310a содержит олигонуклеотид P5 1110, гибридизованный с комплементарной ME-последовательностью (ME′) 1125 и олигонуклеотид P7 1115, гибридизованный с комплементарной ME-последовательностью (ME′) 1125 (то есть, P5:P7); транспозомный комплекс 1310b содержит два олигонуклеотида P5 1110, гибридизованных с комплементарными ME-последовательностями (ME′) 1125 (то есть, P5:P5), а транспозомный комплекс 1310c содержит два олигонуклеотида P7 1117, гибридизованных с комплементарными ME-последовательностями (ME′) 1125 (то есть, P7:P7). Отношения транспозомных комплексов P5:P5, P7:P7 и P5:P7 могут составлять, например, 25:25:50, соответственно.
На фигуре 14 схематически проиллюстрирована диаграмма репрезентативного способа тагментации 1400, проводимого на покрытых транспозомой сферах 1120 как показано на фигуре 13. В этом примере, тагментацию осуществляют путем добавления сферы 1120, содержащей транспозомные комплексы 1310, к раствору ДНК 1410 в буфере для тагментации, а затем ДНК связывают с поверхностью сферы 1120 посредством транспозом 1310. Последовательная тагментация ДНК 1410 приводит к образованию множества мостиковых молекул 1415, расположенных между транспозомами 1310. Длина мостиковых молекул 1415 может зависеть от плотности транспозомных комплексов 1310 на поверхности сферы 1120. В одном из примеров, плотность транспозомных комплексов 1310 на поверхности сферы 1120 может быть скорректирована путем изменения количества олигонуклеотидов P5 и P7, связанных с поверхностью сферы 1120 в стадии 1010 метода 100 как показано на фигуре 10. В другом примере, плотность транспозомных комплексов 1310 на поверхности сферы 1120 может быть скорректирована путем изменения количества комплементарной ME-последовательности (ME′), гибридизованной с олигонуклеотидами P5 и P7 в стадии 1015 метода 1000 как показано на фигуре 10. В другом примере, плотность транспозомных комплексов 1310 на поверхности сферы 1120 может быть скорректирована путем изменения количества фермента транспозазы, добавленного в стадии 1020 метода 1000 как показано на фигуре 1.
Длина мостиковых молекул 1415 не зависит от количества сфер 1120, содержащих связанные с ними транспозомные комплексы 1310 и используемых в реакции тагментации. Аналогичным образом, добавление большего или меньшего количества ДНК 1410 в реакционную смесь для тагментации не приводит к изменению размера конечного тагментированного продукта, но может влиять на выход продукта реакции.
В одном из примеров, сферой 1120 является парамагнитная сфера. В этом примере, очистка продукта реакции тагментации может быть легко достигнута путем иммобилизации сфер 1120 на магните и их последующей промывки. Поэтому, тагментация и последующая ПЦР-амплификация могут быть осуществлены в одном реакционном сосуде («в одном сосуде»).
В одном из своих аспектов, настоящее изобретение относится к способам и композициям, разработанным на основе транспозазы, которые позволяют получить информацию о сцеплении нуклеиновой кислоты-мишени на твердом носителе. В некоторых вариантах осуществления изобретения, указанные композиции и способы позволяют получить информацию о сборке/фазировании. В одном из вариантов осуществления изобретения, твердым носителем является сфера. В одном из вариантов осуществления изобретения, нуклеиновой кислотой-мишенью является ДНК. В одном из вариантов осуществления изобретения, нуклеиновой кислотой-мишенью является геномная ДНК. В некоторых вариантах осуществления изобретения, нуклеиновой кислотой-мишенью является РНК. В некоторых вариантах осуществления изобретения, РНК представляет собой мРНК. В некоторых вариантах осуществления изобретения, нуклеиновой кислотой-мишенью является комплементарная ДНК (кДНК).
В некоторых вариантах осуществления изобретения, транспозоны могут быть иммобилизованы в виде димеров на твердом носителе, таком как сферы, с последующим связыванием транспозазы с транспозонами и с образованием транспозом.
В некоторых вариантах осуществления изобретения, которые, в частности, относятся к образованию транспозом на твердых фазах посредством транспозонов, иммобилизованных на твердой фазе, и добавлению транспозазы, два транспозона могут быть иммобилизованы на твердом носителе в непосредственной близости друг от друга (предпочтительно, на фиксированном расстоянии). Этот метод имеет несколько преимуществ. Во-первых, два транспозона всегда иммобилизуются одновременно, при этом, линкер имеет оптимальную длину, а два этих транспозона имеют оптимальную ориентацию, что способствует эффективному образованию транспозом. Во-вторых, эффективность образования транспозомы не зависит от плотности транспозонов. Два транспозона всегда находятся в правильной ориентации, а расстояние между ними является достаточным для образования транспозом. В-третьих, при рандомизированной иммобилизации транспозонов на поверхностях, транспозоны располагаются на различных расстояниях друг от друга, а поэтому, только одна часть имеет оптимальную ориентацию и оптимальное расстояние, достаточное для эффективного образования транспозом. Следовательно, не все транспозоны превращаются в транспозомы, при этом, могут присутствовать твердофазные иммобилизованные транспозоны, не образующие комплексов. Эти транспозоны могут быть мишенью для транспозиции, поскольку ME-часть представляет собой двухцепочечную ДНК. Это может приводить к снижению эффективности транспозиции или к образованию нежелательных побочных продуктов. Таким образом, транспозомы могут быть получены на твердом носителе, а поэтому они могут быть использованы для получения информации о сцеплении генов посредством тагментации и секвенирования. Репрезентативная схема проиллюстрирована на фигуре 15. В некоторых вариантах осуществления изобретения, транспозоны могут быть иммобилизованы на твердом носителе другими способами, не относящимися к способам химического связывания. Репрезентативными методами иммобилизации транспозонов на твердом носителе могут быть, но не ограничиваются ими, аффинное связывание, такое как связывание «стрептавидин-биотин», «мальтоза - белок, связывающийся с мальтозой», «антиген-антитело», или гибридизация ДНК-ДНК или ДНК-РНК.
В некоторых вариантах осуществления изобретения, транспозомы могут быть подвергнуты предварительной сборке, а затем иммобилизованы на твердом носителе. В некоторых вариантах осуществления изобретения, транспозоны содержат уникальные индексы, штрих-коды и сайты связывания праймеров для амплификации. Транспозаза может быть добавлена в раствор, содержащий транспозоны, с образованием транспозомных димеров, которые могут быть иммобилизованы на твердом носителе. В одном из вариантов осуществления изобретения может быть получено множество наборов сфер, где каждый из этих наборов имеет одинаковый индекс для иммобилизованных транспозонов, в результате чего могут быть получены индексные сферы. К каждой серии индексированных сфер может быть добавлена нуклеиновая кислота-мишень, как показано на фигуре 29A.
В некоторых вариантах осуществления изобретения, к каждой серии индексированных сфер может быть добавлена нуклеиновая кислота-мишень, а затем могут быть проведены отдельные реакции тагментации и ПЦР-амплификации.
В некоторых вариантах осуществления изобретения, нуклеиновая кислота-мишень, индексированные сферы и транспозомы могут быть объединены в виде капелек так, чтобы ряд капелек составляли одну сферу с одной или более молекулами ДНК и адекватными транспозомами.
В некоторых вариантах осуществления изобретения, индексированные сферы могут быть объединены, а затем, в этот пул может быть добавлена нуклеиновая кислота-мишень, после чего может быть проведена реакция тагментации и ПЦР-амплификации в одном реакционном сосуде («в одном сосуде»).
В одном из своих аспектов, настоящее изобретение относится к способам и композициям, применяемым для получения информации о сцеплении генов посредством захвата сцепленных и перенесенных нуклеиновых кислот-мишеней на твердом носителе. В некоторых вариантах осуществления изобретения, транспозицию, сохранаяющую сцепление генов (CPT), осуществляют на ДНК, но эта ДНК должна оставаться интактной (CPT-ДНК), что позволяет получить библиотеки сцепленных нуклеиновых кислот. Информация о сцеплении может быть сохранена с использованием транспозазы для сохранения ассоциации фрагментов матричных нуклеиновых кислот, которые являются смежными в нуклеиновой кислоте-мишени. CPT-ДНК может быть захвачена посредством гибридизации комплементарных олигонуклеотидов, имеющих уникальные индексы или штрих-коды и иммобилизованных на твердом носителе, например, на сферах (фигура 29B). В некоторых вариантах осуществления изобретения, олигонуклеотид, иммобилизованный на твердом носителе, может также включать, помимо штрих-кодов, сайты связывания с праймером и уникальные молекулярные индексы (UMI).
Преимуществом такого использования транспозом для сохранения физической близости фрагментированных нуклеиновых кислот является повышение вероятности того, что фрагментированные нуклеиновые кислоты, происходящие от одной и той же исходной молекулы, например, хромосомы, будут давать одну и ту же информацию по уникальным штрих-кодам и индексам олигонуклеотидов, иммобилизованных на твердом носителе. Это даст возможность получить библиотеку сцепленных секвенирующих нуклеиновых кислот с уникальными штрих-кодами. Эта библиотека сцепленных секвенирующих нуклеиновых кислот может быть секвенирована для получения информации о смежных последовательностях.
На фигурах 16 и 17 схематически представлен репрезентативный вариант вышеуказанного аспекта изобретения, относящегося к получению библиотек сцепленных секвенирующих нуклеиновых кислот, имеющих уникальные штрих-коды или индексы. Этот репрезентативный метод основан на лигировании CPT-ДНК с иммобилизованными олигонуклеотидами на твердом носителе, содержащими уникальные индексы и штрих-коды, и на проведении ПЦР с заменой цепи для получения секвенирующей библиотеки. В одном из вариантов осуществления изобретения, клональные индексированные сферы могут быть получены с использованием иммобилизованных последовательностей ДНК, таких как неспецифический или специфический праймер и индекс. Библиотеки сцепленных нуклеиновых кислот могут быть иммобилизованы на клональных индексированных сферах посредством гибридизации с иммобилизованными олигонуклеотидами с последующим их лигированием. Поскольку внутримолекулярная реакция гибридизации посредством захвата происходит гораздо быстрее, чем межмолекулярая гибридизация, то библиотеки, перенесенные с сохранением сцепления, будут «обертывать» сферы. На фигурах 18 и 19 проиллюстрированы захват CPT-ДНК на клональных индексированных сферах и сохранение информации о сцеплении. ПЦР с заменой цепи позволяет осуществлять перенос информации о клональном индексе сферы на индивидуальную молекулу. Таким образом, каждая библиотека сцепленных нуклеиновых кислот может иметь уникальные индексы.
В некоторых вариантах осуществления изобретения, олигонуклеотид, иммобилизованный на твердом носителе, может содержать частично двухцепочечную структуру, где одна цепь иммобилизована на твердом носителе, а другая цепь частично комплементарна иммобилизованной цепи и образует Y-адаптер. В некоторых вариантах осуществления изобретения, Y-адаптер, иммобилизованный на твердой поверхности, связан с тагментированной сцепленной ДНК посредством лигирования и заполнения брешей, как показано на фигуре 20.
В некоторых вариантах осуществления изобретения, Y-адаптер получают посредством захвата CPT-ДНК с зондом/индексом на твердом носителе, таком как сферы. На фигуре 21 представлена репрезентативная схема получения таких Y-адаптеров. Использование этих Y-адаптеров дает уверенность, что каждый фрагмент может стать секвенирующей библиотекой. Это позволяет охватить большую область секвенирования.
В некоторых вариантах осуществления изобретения, свободные транспозомы могут быть отделены от CPT-ДНК. В некоторых вариантах осуществления изобретения, такое отделение свободных транспозом осуществляют посредством эксклюзионной хроматографии. В одном из вариантов осуществления изобретения, такое отделение может быть достигнуто на колонках MicroSpin S-400 HR (GE Healthcare Life Sciences, Pittsburgh, PA). На фигуре 22 проиллюстрирован электрофорез в агарозном геле для CPT-ДНК, отделенных от свободных транспозом.
Захват сцепленных и перенесенных нуклеиновых кислот-мишеней на твердом носителе посредством гибридизации имеет ряд уникальных преимуществ. Во-первых, этот метод основан на гибридизации, но не на транспозиции. Степень внутримолекулярной гибридизация выше степени межмолекулярной гибридизации. Таким образом, шансы получения библиотек со сцепленными и перенесенными нуклеиновыми кислотами на одной молекуле ДНК-мишени для обертывания уникальной индексированной сферы гораздо выше, чем шансы получения библиотек на двух или более других отдельных молекулах ДНК-мишени для обертывания уникальной индексированной сферы. Во-вторых, транспозиция ДНК и штриховое кодирование перенесенной ДНК могут быть осуществлены в две отдельных стадии. В-третьих, можно решить проблемы, связанные со сборкой активных транспозом на сферах и с оптимизацией плотности транспозонов на твердых поверхностях. В-четвертых, продукты аутотранспозиции могут быть удалены путем колоночной очистки. В-пятых, поскольку перенесенная и сцепленная ДНК содержит бреши, то такая ДНК является более гибкой, что будет снижать нагрузку на плотность транспозиции (размер вставок) по сравнению с плотностью, достигаемой с применением методов иммобилизации транспозомы на сферах. В-шестых, в этом методе могут быть использованы схемы комбинаторного штрих-кодирования. В-седьмых, может быть облегчена процедура ковалентного связывания индексированных олигонуклеотидов со сферами. Таким образом, уменьшается вероятность замены индекса. В-восьмых, реакция тагментации и последующая реакция ПЦР-амплификации могут быть мультиплексными и могут быть проведены в одном реакционном сосуде («в одном сосуде»), что освобождает от необходимости проведения отдельных реакций для каждых индексных последовательностей.
В некоторых вариантах осуществления изобретения, множество уникальных штрих-кодов для всех нуклеиновых кислот-мишеней может быть встроено во время транспозиии. В некоторых вариантах осуществления изобретения, каждый штрих-код включает первую последовательность со штрих-кодом и вторую последовательность со штрих-кодом, имеющие сайт фрагментации, расположенные между ними. Первая последовательность со штрих-кодом и вторая последовательность со штрих-кодом могут быть идентифицированы или сконструированы так, чтобы они спаривались друг с другом. Спаривание может носить информативный характер, а поэтому первый штрих-код должен быть ассоциирован со вторым штрих-кодом. Спаренные последовательности со штрих-кодом могут быть преимущественно использованы для сбора данных по секвенированию библиотеки матричных нуклеиновых кислот. Так, например, идентификация первой матричной нуклеиновой кислоты, содержащей первую последовательность со штрих-кодом, и второй матричной нуклеиновой кислоты, содержащей вторую последовательность со штрих-кодом, где указанная последовательность спарена с первой последовательностью, показала, что первая и вторая матричные нуклеиновые кислоты представляют собой последовательности, смежные друг с другом в нуклеиновой кислоте-мишени. Такие методы могут быть применены для сборки репрезентативных последовательностей нуклеиновой кислоти-мишени de novo, где такая сборки не требует эталонного генома.
В одном из своих аспектов, настоящее изобретение относится к способам и композициям, применяемым для получения библиотеки последовательностей специфического фрагмента ДНК с использованием «дробовика».
В одном из вариантов осуществления изобретения, клональные индексированные сферы получают с использованием иммобилизованных олигонуклеотидных последовательностей: неспецифических или специфических праймеров и уникальных индексов. Нуклеиновую кислоту-мишень добавляют к клональным индексированным сферам. В некоторых вариантах осуществления изобретения, нуклеиновой кислотой-мишенью является ДНК. В одном из вариантов осуществления изобретения, ДНК-мишень является денатурированной. ДНК-мишень гибридизуется сначала с праймерами, содержащими уникальные индексы, иммобилизованные на твердой поверхности (например, на сферах), а затем с другими праймерами, имеющими тот же самый индекс. Праймеры на сферах амплифицируют ДНК. При этом, могут быть проведены один или более дополнительных раундов амплификации. В одном из вариантов осуществления изобретения, амплификация может быть осуществлена для целого генома с использованием иммобилизованных на сфере праймеров, имеющих 3'-рандомизированную n-мерную последовательность. В предпочтительном варианте осуществления изобретения, рандомизированный n-мер содержит псевдокомплементарные основания (2-тиотимин, 2-амино-dA, N4-этилцитозин и т.п.), что предотвращает взаимодействия праймера с праймером в процессе амплификации (Hoshika, S; Chen, F; Leal, NA; Benner, SA, Angew. Chem. Int. Ed. 49(32) 5554-5557 (2010). На фигуре 23 представлена репрезентативная схема получения библиотеки последовательностей специфического фрагмента ДНК с использованием «дробовика». Может быть генерирована клональная индексированная секвенирующая библиотека, которая может представлять собой библиотеку амплифицированных продуктов. В одном из вариантов осуществления изобретения, такая библиотека может быть получена путем транспозиции. Информация о последовательности клональной индексированной библиотеки может быть использована для сбора информации о смежных последовательностях по данным об индексах, служащих в качестве ориентира. На фигуре 24 представлена репрезентативная схема сборки информации о последовательности клональной индексированной секвенирующей библиотеки.
Способы согласно вышеуказанным вариантам изобретения имеют несколько преимуществ. Внутримолекулярная амплификация на сферах происходит гораздо быстрее, чем межмолекулярная амплификация. Таким образом, продукты на сферах имеют один и тот же индекс. Библиотека специфического фрагмента ДНК может быть получена методом «дробовика». Рандомизированные праймеры амплифицируют матрицу в произвольных положениях, а поэтому библиотека, полученная методом «дробовика» и имеющая тот же самый индекс, может быть генерирована из специфической молекулы, а информация о последовательности может быть собрана с использованием индексированной последовательности. Значительное преимущество способов согласно вышеуказанным вариантам изобретения заключается в том, что реакции могут быть мультиплексными и проводиться за одну реакцию (в одном реакционном сосуде), и для их проведения не требуется множества отдельных лунок. Многие клональные индексированные сферы могут быть получены так, чтобы множество различных фрагментов могло иметь уникальные метки, что позволило бы дифференцировать родительские аллели для одних и тех же геномных областей. При высоком числе индексов, вероятность того, что копия ДНК отца и копия ДНК матери будут иметь один и тот же индекс для одной и той же геномной области, довольно мала. Этот способ имеет то преимущество, что внутренние реакции проходят гораздо быстрее, чем внешние, а поэтому сферы будут, в основном, давать фактическое распределение в более широком физическом пространстве.
В некоторых вариантах всех вышеуказанных аспектов изобретения, этот способ может быть применен для внеклеточных ДНК (свободной ДНК или свДНК) в анализах на свДНК. В некоторых вариантах осуществления изобретения, свДНК получают из плазмы и плацентарной жидкости.
В одном из вариантов осуществления изобретения, плазма может быть выделена из неразведенной цельной крови с использованием мембранного сепаратора плазмы, работающего на основе седиментации (Liu et al. Anal Chem. 2013 Nov. 5;85(21):10463-70). В одном из вариантов осуществления изобретения, зона сбора плазмы сепаратора может содержать твердый носитель, включающий транспозомы. Твердый носитель, включающий транспозомы, может захватывать свДНК из выделенной плазмы по мере ее отделения от цельной крови и может концентрировать свДНК и/или тагментировать ДНК. В некоторых вариантах осуществления изобретения, тагментация также позволяет вводить уникальные штрих-коды для последующего разуплотнения после секвенирования пула библиотек.
В некоторых вариантах осуществления изобретения, зона сбора сепаратора может содержать маточную ПЦР-смесь (праймеры, нуклеотиды, буферы, металлы) и полимеразу. В одном из вариантов осуществления изобретения, маточная смесь может использоваться в сухой форме так, чтобы ее можно было развести по мере выхода плазмы из сепаратора. В некоторых вариантах осуществления изобретения, праймерами являются рандомизированные праймеры. В некоторых вариантах осуществления изобретения, праймеры могут быть специфичными к конкретному гену. ПЦР-амплификация свДНК может приводить к получению библиотеки непосредственно из выделенной плазмы.
В некоторых вариантах осуществления изобретения, зона сбора сепаратора может содержать маточную ОТ-ПЦР-смесь (праймеры, нуклеотиды, буферы, металлы), обратную транскриптазу и полимеразу. В некоторых вариантах осуществления изобретения, праймерами являются рандомизированные праймеры или олиго-dT-праймеры. В некоторых вариантах осуществления изобретения, праймеры могут быть специфичными к конкретному гену. Полученная кДНК может быть использована для секвенирования. Альтернативно, кДНК может быть обработана транспозомами, иммобилизованными на твердом носителе, для получения библиотеки последовательностей.
В некоторых вариантах осуществления изобретения, сепаратор плазмы может включать штрих-коды (штрих-коды 1D или 2D). В некоторых вариантах осуществления изобретения, аппарат для сепарации может включать устройство для забора крови. Это позволяет осуществлять прямую доставку крови в сепаратор плазмы и в устройство для получения библиотеки. В некоторых вариантах осуществления изобретения, устройство может включать нижерасположенный анализатор последовательности. В некоторых вариантах осуществления изобретения, анализатором последовательности является одноразовый секвенатор. В некоторых вариантах осуществления изобретения, секвенатор позволяет установить очередность взятия образцов перед их серийным секвенированием. Альтернативно, секвенатор может обеспечивать рандомизированный доступ образцов в зону их секвенирования.
В некоторых вариантах осуществления изобретения, зона сбора плазмы может содержать субстраты на основе двуокиси кремния для концентрирования неклеточной ДНК.
Одновременное фазирование и детектирование метилирования
5-метилцитозин (5-Me-C) и 5-гидроксиметилцитозин (5-гидрокси-С) также известны как эпи-модификации, играющие важную соль в метаболизме и дифференцировке клеток, а также в развитии рака. Авторами настоящего изобретения было неожиданно обнаружено, что фазирование и одновременное детектирование метилирования может быть осуществлено с применением способов и композиций согласно изобретению. Способы согласно изобретению позволяют одновременно осуществлять CPT-seq на сферах (с индексированными сцепленными библиотеками) и детектирование метилирования ДНК. Так, например, отдельные библиотеки, полученные на сферах, могут быть обработаны бисульфитом с последующим превращением неметилированных, но не метилированных, цитозинов (C) в U, что позволяет детектировать 5-Me-C. С помощью дополнительного анализа на фазирование с использованием гетерозиготных SNP могут быть получены эпи-модификацию-фазирующие блоки, имеющие размер в пределах мультимегаоснований.
В некоторых вариантах осуществления изобретения, размер анализируемой ДНК может составлять приблизительно от 100 оснований до мультимегаоснований. В некоторых вариантах осуществления изобретения, размер анализируемой ДНК может составлять приблизительно 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 1200, 1300, 1500, 2000, 3000, 3500, 4000, 4500, 5000, 5500, 6000, 6500, 7000, 7500, 8000, 8500, 9000, 9500, 10000, 10500, 11000, 11500, 12000, 12500, 13000, 14000, 14500, 15000, 15500, 16000, 16500, 17000, 17500, 18000, 18500, 19000, 19500, 20000, 20500, 21000, 21500, 22000, 22500, 23000, 23500, 24000, 24500, 25000, 25500, 26000, 26500, 27000, 27500, 28000, 28500, 29500, 30000, 30500, 31000, 31500, 32000, 33000, 34000, 35000, 36000, 37000, 38000, 39000, 40000, 42000, 45000, 50000, 55000, 60000, 65000, 70000, 75000, 80000, 85000, 90000, 95000, 100000, 110000, 120000, 130000, 140000, 150000, 160000, 170000, 180000, 200000, 225000, 250000, 300000, 350000, 400000, 450000, 500000, 550000, 600000, 650000, 700000, 750000, 800000, 850000, 900000, 1000000, 1250000, 1500000, 2000000, 2500000, 3000000, 4000000, 5000000, 6000000, 7000000, 8000000, 9000000, 10000000, 15000000, 20000000, 30000000, 40000000, 50000000, 75000000, 100000000 или более оснований.
Другие эпи-модификации, такие как 5-гидрокси-C, продукты окисления ДНК, продукты алкилирования ДНК, футпринтинг на гистоне и т.д., также могут быть проанализированы на фазирование с применением описанных способов и композиций согласно изобретению.
В некоторых вариантах осуществления изобретения, ДНК сначала превращают в сцепленные с индексом библиотеки на твердом носителе. Отдельные индексированные библиотеки, размер которых гораздо меньше, чем размер исходной ДНК, менее предрасположены к фрагментации, поскольку такие отдельные библиотеки имеют меньший размер. Даже при потере небольшой фракции индексированных библиотек, информация о фазировании еще сохраняется для всего диапазона индексированной молекулы ДНК. Так, например, если 100 т.п.о.-молекула, полученная путем традиционного превращения под действием бисульфита (BSC), была фрагментирована наполовину, то сцепление ограничено 50 т.п.о. В описанных здесь способах, сначала индексируют 100 т.п.о.-библиотеку и даже в случае потери части отдельных библиотек, сцепление еще будет составлять ~100 т.п.о. (за исключением очень маловероятного события, когда все потерянные библиотеки находятся у одного конца молекулы ДНК). Кроме того, способы, описанные в настоящей заявке, имеют дополнительные преимущества, заключающиеся в том, что при проведении этих способов не требуются дополнительные стадии очистки по сравнению с традиционными методами превращения под действием бисульфита, что также способствует повышению выхода. В способах согласно изобретению, сферы, после их превращения под действием бисульфита, просто промывают. Кроме того, поскольку ДНК связана с твердой фазой, то может быть легко осуществлен буферный обмен с минимальной потерей ДНК (индексированных библиотек) и уменьшением времени на обработку.
Репрезентативная схема одновременного фазирования и детектирования метилирования представлены на фиг. 43. Технологическая схема такого процесса состоит из тагментации ДНК на сферах; лигирования областей-повторов размером 9 п.о. с заполнением брешей; удаления Tn5 посредством ДСН; и превращения отдельных библиотек на сферах под действием бисульфита. Превращение под действием бисульфита осуществляют в условиях денатурации для гарантии того, что соседние комплементарные библиотеки не будут подвергаться повторному отжигу, который снижает эффективность превращения под действием бисульфита. BCS превращает неметилированные C в U, а метилированные C не подвергаются такому превращению.
На фиг. 44 представлена альтернативная репрезентативная схема одновременного фазирования и детектирования метилирования. После получения секвенирующих библиотек посредством транспозиции, часть библиотек, лигированных с заполнением брешей, разлагается с образованием одноцепочечных матриц. Для превращения под действием бисульфита, одноцепочечные матрицы требуют более мягких условий, поскольку эти матрициы уже являются одноцепочечными, что снижает потери библиотек или повышает эффективность превращения под действием бисульфита. В одном из вариантов осуществления изобретения, смесь 3'-тио-защищенных транспозонов (Exo-резистентных) и незащищенных транспозонов используют на одной и той же сфере. Ферменты, например, Exo I, могут быть применены для расщепления библиотек, не защищенных тио-группой, что способствует превращению этих библиотек в одноцепочечные библиотеки. С использованием смеси тио-защищенных транспозонов/незащищенных транспозонов (50:50), 50% библиотек превращаются в одноцепочечные библиотеки (50% библиотек имеют один защищенный и один незащищенный транспозон, то есть, комплементарную цепь), 25% вообще не подвергались превращению (то есть, оба транспозона были тио-защищенными), а 25% подвергались превращению с удалением всей библиотеки (оба транспозона были не защищены).
Одним из недостатков метода бисульфитного превращения ДНК, связанной с твердой фазой, такой как стрептавидиновые магнитные сферы, является длительная обработка связанной со сферами ДНК бисульфитом натрия при высоких температурах, которая приводит к разрушению как ДНК, так и сфер. Для снижения степени повреждения ДНК, в реакционную смесь, до ее обработки бисульфитом, добавляют носитель ДНК (то есть, лямбда ДНК). Было установлено, что даже в присутствии носителя ДНК теряется приблизительно 80% исходной ДНК. В результате этого, блоки сцепления CPTSeq имеют меньшее число членов, чем это наблюдается в протоколе проведении традиционного CPTSeq.
Поэтому, для увеличения выхода ДНК, в предложенный здесь протокол проведения Epi-CPTSeq добавляют несколько стратегий. Первая стратегия основана на снижении размера вставок в библиотеку путем введения более плотной совокупности транспозомных комплексов, связанных со стрептавидиновыми сферами. При снижении размера библиотеки, меньшее число элементов библиотеки будет подвергаться разрушению после обработки бисульфитом.
Второй стратегией увеличения выхода ДНК в соответствии с протоколом Epi-CPTSeq является ферментативное восстановление разрушенных элементов библиотеки. Целью такой стратегии восстановления является повторное присоединение общей 3'-последовательности, необходимой для амплификации библиотеки, к элементам библиотеки, связанным со сферами, поскольку эти элементы подвергались расщеплению и теряли свою 3'-часть в процессе обработки бисульфитом. После присоединения общей 3'-последовательности, эти элементы снова могут быть подвергнуты ПЦР-амплификации и секвенированию. На фигурах 67 и 68 представлена репрезентативная схема этой стратегии. Двухцепочечные элементы библиотеки CPTSeq были денатурированы и подвергнуты бисульфитному превращению (верхняя панель). В процессе бисульфитного превращения, одна из цепей ДНК была повреждена (средняя панель), что приводило к потере общей ПЦР-последовательности на 3'-конце. Стратегии «спасения» матрицы способствуют восстановлению общей 3'-последовательности (показанной зеленым), необходимой для ПЦР-амплификации (нижняя панель). В одном из примеров используются концевая трансфераза в присутствии 3'-фосфорилированного олиго-аттенюатора; последовательность, содержащая секвенирующий адаптер, а затем, олиго-dT-фрагмент (фигура 68A). Вкратце, TdT присоединяет фрагмент из 10-15 dA к 3'-концу разрушенного элемента библиотеки, что приводит к гибридизации с олиго-dT-частью олиго-аттенюатора. Образование этой гибридной ДНК приводит к прекращению реакции TdT и к образованию матрицы для последующего удлинения 3'-конца разрушенного элемента библиотеки под действием ДНК-полимеразы.
В альтернативной технологической схеме (фигура 68B), реакцию присоединения TdT-хвоста осуществляют в присутствии частично двухцепочечного олиго-аттенюатора, содержащего одноцепочечную олиго-dT-часть и 5'-фосфорилированную двухцепочечную секвенирующую часть адаптера. После прекращения реакции TdT, разрыв между последним добавлением dA и 5'-фосфорилированным олиго-аттенюатором репарируют с помощью ДНК-лигазы.
Обе описанные технологические схемы основаны на недавно разработанной регулируемой реакции присоединения TdT-хвоста, описанной в публикации заявки на патент США 20150087027. Общий секвенирующий адаптер может быть также присоединен к 3'-концу разрушенных элементов библиотеки путем только что встроенной оцДНК-матрицы, переключающей активность MMLV-RT. Короче говоря, MMLV RT и матрицу, переключающие олигонуклеотид (TS_oligo), добавляют к разрушенной ДНК (фигура 68C). В первой стадии этой реакции, обратная транскриптаза добавляет несколько дополнительных нуклеотидов к 3'-концам одноцепочечного фрагмента ДНК, и эти пары оснований с олиго (N)-последовательностью присутствуют у 3'-конца одного из TS_oligo. Затем, матрица на основе обратной транскриптазы, переключающая активность, добавляет последовательности общих гибридизованных праймеров к 3'-концу BSC-разрушенного элемента библиотеки, что приводит к восстановлению способности к ПЦР-амплификации в присутствии общих секвенирующих праймеров.
При проведении третьей стратегии, для «спасения» элементов библиотеки, потерявших свои общие последовательности у 3'-конца в процессе бисульфитного превращения, может быть применен набор Epicentre's EpiGenome для осуществления метода конструирования библиотеки «после бисульфитного превращения». Как показано на фигуре 69, этот метод «спасения» библиотеки проводят с применением 3'-фосфорилированных олигонуклеотидов с общими последовательностями, за которыми расположен короткий фрагмент рандомизированной последовательности. Эти короткие рандомизированные последовательности гибридизуются с одноцепочечной ДНК, обработанной бисульфитом, а общие последовательности затем копируют на разрушенную цепь библиотеки с помощью ДНК-полимеразы.
На фигуре 74 проиллюстрировна четвертая стратегия усовершентсвования методов секвенирования на сферах путем обработки бисульфитом. Первая общая последовательность, содержащая метку для захвата, ковалентно присоединена к 5'-концам ДНК. Первая общая последовательность может быть присоединена к ДНК различными методами, включая одностороннюю транспозицию (как показано на фигуре), лигирование адаптера или лигирование адаптера под действием концевой трансферазы (TdT) как описано в публикации заявки на патент США 20150087027.
Затем, ДНК денатурируют (например, путем инкубирования при высокой температуре) и присоединяют к твердому носителю. Если в качестве метки для захвата на CS1 служит биотин, то, например, ДНК может быть связана с применением стрептавидиновых магнитных сфер (как показано на фигуре). После присоединения к твердому носителю может быть легко осуществлен буферный обмен.
В следующей стадии осуществляют бисульфитное превращение оцДНК. ДНК, если она присутствует в одноцепочечной форме, должна быть легко доступна для бисульфитного превращения, и эффективность ее превращения до 95% может быть достигнута с помощью модифицированного варианта набора Promega's Methyl Edge BSC (фигура 75).
После бисульфитного превращения, вторую общую последовательность ковалентно присоединяют к 3'-концу оцДНК, связанной с твердым носителем. Для ковалентного присоединения олигонуклеотидов к оцДНК были применены несколько методов, описанных выше. С примененеием метода лигирования аттенюатора/адаптера TdT, эффективность лигирования может составлять >95%. В результате, конечные выходы библиотеки, полученные с применением предложенной технологической схемы MethylSeq, должны превышать выходы, полученные с применением уже существующих методов.
В конечной стадии осуществляют ПЦР для амплификации библиотеки и ее удаления из твердого носителя. ПЦР-праймеры могут быть сконструированы так, чтобы они присоединяли дополнительные общие последовательности, такие как секвенирующие адаптеры, к концам библиотеки MethylSeq.
Получение библиотек различных размеров в одном анализе
Точность сборки геномов зависит от применения технологий масштабирования различных длин. Так, например, все методы «дробовика» (100 п.о.), то есть, методы спаривания (~3 т.п.о.) с -Hi-C (в масштабе мегаоснований) представляют собой методы, которые применяют в целях последовательного улучшения сборки и увеличения длин контигов. Недостаток этого метода заключается в том, что для его осуществления необходимо проводить множество анализов, а поэтому такой многостадийный метод является очень трудоемким и дорогостоящим. Описанные здесь композиции и способы могут быть применены к различным шкалам длин в одном анализе.
В некоторых вариантах осуществления изобретения, получение библиотеки может быть осуществлено в одном анализе с использованием твердого носителя различных размеров, например, сфер. Размер каждой сферы позволяет определить размер или интервал размеров специфической библиотеки и физический размер сферы, определяющей размер библиотеки. Все сферы различных размеров имеют уникальные клональные индексы, которые переносят в библиотеку. Так, например, библиотеки различных размеров получают с использованием каждой библиотеки с различными длинами, имеющими уникальный индекс. Библиотеки с различными длинами получают одновременно в одном и том же физическом пространстве, что позволяет снизить стоимость технологического процесса и усовершенствовать весь этот процесс. В некоторых вариантах осуществления изобретения, специфический твердый носитель каждого конкретного размера, например, сфера конкретного размера имеет уникальный индекс. В некоторых других вариантах осуществления изобретения, множество различных индексов твердых носителей одного и того же размера, например, сфер, также получают так, чтобы множество молекул ДНК могли иметь индексы, распределенные по интервалу данных значений. На фиг. 45 представлена репрезентативная схема создания библиотеки различных размеров с применением клональных индексированных сфер различных размеров в одном анализе.
В некоторых вариантах осуществления изобретения, размер генерируемых библиотек составляет приблизительно 50, 75, 100, 150, 200, 250, 300, 350, 400, 500, 600, 700, 800, 900, 1000, 1200, 1300, 1500, 2000, 3000, 3500, 4000, 4500, 5000, 5500, 6000, 6500, 7000, 7500, 8000, 8500, 9000, 9500, 10000, 10500, 11000, 11500, 12000, 12500, 13000, 14000, 14500, 15000, 15500, 16000, 16500, 17000, 17500, 18000, 18500, 19000, 19500, 20000, 20500, 21000, 21500, 22000, 22500, 23000, 23500, 24000, 24500, 25000, 25500, 26000, 26500, 27000, 27500, 28000, 28500, 29500, 30000, 30500, 31000, 31500, 32000, 33000, 34000, 35000, 36000, 37000, 38000, 39000, 40000, 42000, 45000, 50000, 55000, 60000, 65000, 70000, 75000, 80000, 85000, 90000, 95000, 100000, 110000, 120000, 130000, 140000, 150000, 160000, 170000, 180000, 200000, 225000, 250000, 300000, 350000, 400000, 450000, 500000, 550000, 600000, 650000, 700000, 750000, 800000, 850000, 900000, 1000000, 1250000, 15000000, 2000000, 2500000, 3000000, 4000000, 5000000, 6000000, 7000000, 8000000, 9000000, 10000000, 15000000, 20000000, 30000000, 40000000, 50000000, 75000000, 100000000 или более оснований.
В некоторых вариантах осуществления изобретения, обсуждаемые выше библиотеки, имеющие длины различных масштабов, могут быть использованы для сборки псевдогенов, паралогов и т.п. вместо библиотеки, имеющей длину одного большого масштаба. В некоторых вариантах осуществления изобретения, библиотеки, имеющие длины различных масштабов, получают одновременно в одном анализе. Преимущество этого метода состоит в том, что по меньшей мере одна шкала длин имеет уникальную область, которая охватывает только псевдоген и/или ген, но не то и другое. Так, например, вариантам, детектируемым по этой шкале длин, может быть присвоен уникальный вариант, по которому можно определить, является ли он геном или псевдогеном. То же самое правило справедливо и для вариантов различных копий, паралогов и т.п. Точность сборки зависит от применения различных шкал длин. С применением описанных здесь методов могут быть получены сцепленные с индексами библиотеки различных длин в одном анализе вместо отдельных различных библиотек с различными длинами. На фиг. 46 представлена репрезентативная схема определения генетических вариантов с применением библиотек с различными шкалами длин.
Анализ геномных вариантов
Описанные здесь композиции и способы относятся к анализам геномных вариантов. Репрезентативными геномными вариантами являются, но не ограничиваются ими, делеции, межхромосомные транслокации, дупликации, паралоги, межхромосомные сцепления генов. В некоторых вариантах осуществления изобретения, описанные здесь композиции и способы применяются для получения информации о фазировании геномных вариантов. В представленной ниже таблице проиллюстрированы репрезентативные межхромосомные сцепления генов.
Таблица 1: Межхромосомные сцепления генов
В таблице 2 представлены репрезентативные делеции, присутствующие в хромосоме 1
Таблица 2: Репрезентативные делеции в хромосоме 1
В некоторых вариантах осуществления изобретения, нуклеиновая кислота-мишень может быть фрагментирована до ее обработки транспозомами. Репрезентативными методами фрагментации являются, но не ограничиваются ими, обработка ультразвуком, механический сдвиг и расщепление рестриктирующими ферментами. Фрагментирование нуклеиновой кислоты-мишени до тагментации (фрагментирования и мечения) применяется преимущественно для сборки/фазирования псевдогенов (например, CYP2D6). Длинные «островки» (>30 т.п.о.) индексированных сцепленных ридов охватывают псевдогены A и A' как показано на фигуре 64. Из-за высокой гомологии последовательностей трудно определить, какой из вариантов принадлежит гену A, а какой гену A'. Более короткие варианты будут связывать один вариант псевдогенов с уникальными соседними последовательностями. Такие более короткие островки могут быть получены путем фрагментирования нуклеиновой кислоты-мишени до тагментации.
Сцепленные транспозомы
В некоторых вариантах осуществления изобретения, транспозазы являются мультимерными в транспозомном комплексе, например, они образуют димеры, тетрамеры и т.п. в транспозомном комплексе. Авторами настоящего изобретения было неожиданно обнаружено, что сцепление мономерных транспозаз в мультимерном транспозомном комплексе или сцепление транспозонных концов транспозомного мономера в мультимерном транспозомном комплексе имеет несколько преимуществ. Во-первых, сцепление транспозаз или транспозонов приводит к образованию комплексов, которые являются более стабильными и представляют крупную фракцию в активном состоянии. Во-вторых, более низкие концентрации транспозом могут быть использованы для фрагментирования посредством реакции транспозиции. В-третьих, сцепление приводит к уменьшению числа замен мозаичных концов (ME) транспозомных комплексов, и тем самым к уменьшению вероятности смешивания штрих-кодов или молекул адаптера. Такая замена ME-концов возможна в том случае, когда комплексы расположены на определенном расстоянии друг от друга и преобразованы, или в том случае, когда транспозомы иммобилизованы на твердом носителе посредством стрептавидина/биотина, если взаимодействие стрептавидина/биотина может нарушаться и восстанавливаться, или если наблюдается контаминация. Авторы настоящего изобретения отмечают, что значительная модификация или замена ME-концов наблюдается в различных реакционных условиях. В некоторых вариантах осуществления изобретения, замена может составлять до 15%. Процент замен увеличивается при высокой концентрации солевого буфера и снижается в глутаматном буфере. На фигурах 57 и 58 представлены некоторые возможные механизмы замены ME.
В некоторых вариантах осуществления изобретения, субъединицы транспозазы в транспозомном комплексе могут быть связаны друг с другом ковалентными и нековалентными связями. В некоторых вариантах осуществления изобретения, мономеры транспозазы могут быть связаны до получения транспозомного комплекса (перед добавлением транспозонов). В некоторых вариантах осуществления изобретения, мономеры транспозазы могут связываться после образования транспозомы.
В некоторых вариантах осуществления изобретения, нативные аминокислотные остатки могут быть заменены аминокислотой цистеином (Cys) в мультимерной пограничной области для стимуляции образования дисульфидной связи. Так, например, в транспозазе Tn5, Asp468, Tyr407, Asp461, Lys459, Ser458, Gly462, Ala466, Met470 могут быть заменены Cys для стимуляции образования дисульфидной связи между мономерными субъединицами, как показано на фигурах 59 и 60. Для транспозазы Mos-1, репрезентативными аминокислотам, которые могут быть заменены цистеином, являются, но не ограничиваются ими, Leu21, Leu32, Ala35, His20, Phe17, Phe36, Ile16, Thr13, Arg12, Gln10, Glu9 как показано на фиг. 61. В некоторых вариантах осуществления изобретения, модифицированные транспозазы, в которых аминокислотные остатки заменены цистеином, могут быть химически связаны друг с другом перекрестными связями под действием химического перекрестно-сшивающего линкера посредством малеимидных или пиридилдитиоловых реакционноспособных групп. Репрезентативные химические перекрестно-сшивающие линкеры являются коммерчески доступными и поставляются компаниями Pierce Protein Biology/ThermoFisher Scientific (Grand Island, NY, USA).
В некоторых вариантах осуществления изобретения, транспозомные мультимерные комплексы могут быть ковалентно связаны с твердым носителем. Репрезентативными твердыми носителями являются, но не ограничиваются ими, наночастицы, сферы, поверхности проточной кюветы, колоночные матрицы. В некоторых вариантах осуществления изобретения, твердые поверхности могут быть покрыты аминогруппами. Модифицированная транспозаза, в которой аминокислотные остатки были заменены цистеином, может быть подвергнута химической реакции перекрестного связывания с такими аминогруппами посредством перекрестно-сшивающего линкера, связываюшего амин с сульфгидрилом (то есть, сукцинимидил-4-(N-малеимидометил)циклогексан-1-карбоксилата (SMCC)). Репрезентативная схема представлена на фигуре 62. В некоторых вариантах осуществления изобретения, перекрестно-сшивающий линкер, такой как малеимид-ПЭГ-биотин, может быть использован для присоединения dTnp к твердой поверхности, покрытой стрептавидином.
В некоторых вариантах осуществления изобретения, ген транспозазы может быть модифицирован для экспрессии мультимерного белка в одном полипептиде. Так, например, гены Tn5 или Mos-1 могут быть модифицированы для экспрессии двух белков Tn5 или Mos-1 в одном полипептиде. Аналогичным образом, ген транспозазы Mu может быть модифицирован так, чтобы он кодировал четыре единицы транспозазы mu в одном полипептиде.
В некоторых вариантах осуществления изобретения, транспозонные концы транспозомной мономерной единицы могут быть сцеплены с образованием сцепленного транспозомного мультимерного комплекса. Сцепление транспозонных концов позволяет встраивать сайты праймеров, секвенирующие праймеры, праймеры для амплификации или определить, какую роль может играть ДНК в образовании гДНК без фрагментирования ДНК-мишени. Встраивание таких функциональных групп имеет определенные преимущества при проведении анализов на гаплотипы или анализов на мечение области стыка, где необходимо получить информацию об интактных молекулах, или где важное значение имеет взятие части образцов из пробы. В некоторых вариантах осуществления изобретения, транспозонные концы транспозом Mu могут быть сцеплены с транспозазой Mu/транспозоном, имеющими конфигурацию типа «петли». Поскольку Mu является тетрамером, то возможны различные конфигурации, включая, но не ограничиваясь ими, сцепление R2UJ и/или R1UJ с R2J и/или R1J. При таких конфигурациях, R2UJ и R1UJ могут быть сцеплены, а могут быть и не сцеплены с R2J и R1J, соответственно. На фигуре 63 представлен транспозомный комплекс Mu, в котором транспозонные концы являются сцепленными. В некоторых вариантах осуществления изобретения, транспозонные концы Tn5 или транспозонные концы транспозом Mos1 могут быть сцепленными.
Используемый здесь термин «транспозон» означает двухцепочечную ДНК, которая имеет только нуклеотидные последовательности (последовательности «транспозонных концов»), необходимые для образования комплекса с ферментом транспозазой или интегразой, которые являются функциональными в реакции транспозиции in vitro. Транспозон образует «комплекс» или «синаптический комплекс» или «транспозомный комплекс» или «транспозомную композицию», содержащую транспозазу или интегразу, которая распознает транспозон и связывается с транспозоном, где указанный комплекс обладает способностью встраивать или переносить транспозон в ДНК-мишень, с которой его инкубируют в реакции транспозиции in vitro. Транспозон имеет две комплементарные последовательности, состоящие из «перенесенной последовательности транспозона» или «перенесенной цепи» и «неперенесенной последовательности транспозона» или «неперенесенной цепи». Так, например, один транспозон, образующий комплекс с гиперактивной транспозазой Tn5 (например, EZ-Tn5™ Transposase, EPICENTRE Biotechnologies, Madison, Wis., USA), которая является активной в реакции транспозиции in vitro, содержит перенесенную цепь, имеющую нижеследующую «перенесенную транспозонную последовательность»:
5' AGATGTGTATAAGAGACAG 3'
и неперенесенную цепь, имеющую нижеследующую «неперенесенную транспозонную последовательность»:
5' CTGTCTCTTATACACATCT 3'.
3'-конец перенесенной цепи присоединяют к ДНК-мишени или переносят в ДНК-мишень посредством реакции транспозиции in vitro. Неперенесенную цепь, имеющую транспозонную последовательность, комплементарную перенесенной последовательности транспозонного конца, не присоединяют к ДНК-мишени или не переносят в ДНК-мишень посредством реакции транспозиции in vitro. В некоторых вариантах осуществления изобретения, транспозонные последовательности могут содержать одну или более из таких последовательностей, как штрих-код, последовательность адаптера, последовательность метки, последовательность, связывающуюся с праймером, последовательность для захвата и уникальную последовательность молекулярного идентификатора (UMI).
Используемый здесь термин «адаптер» означает последовательность нуклеиновой кислоты, которая может содержать штрих-код; последовательность, связывающуюся с праймером; последовательность для захвата; последовательность, комплементарную последовательности для захвата; уникальную последовательность молекулярного идентификатора (UMI); аффинную группу и рестрикционный сайт.
Используемый здесь термин «информация о сцеплении» означает пространственную взаимосвязь между двумя или более фрагментами ДНК, выявленную исходя из общей информации. Общий аспект такой информации может относиться к взаимодействиям смежных фрагментов, отдельных фрагментов и фрагментов, находящихся на пространственном расстоянии друг от друга. Информация, относящаяся к этим взаимодействиям, в свою очередь, облегчает иерархическую сборку и мечения ридов последовательности, происходящих от фрагментов ДНК. Такая информация о сцеплении повышает эффективность и точность такой сборки или картирования, поскольку традиционные методы сборки или картирования, применяемые в комбинации со стандартным секвенированием методом «дробовика», не позволяют точно определить родственные геномные ориджины или координаты ридов отдельных последовательностей, так как они находятся в пространственной взаимосвязи между двумя или более фрагментами ДНК, от которых происходят риды отдельных последовательностей. Поэтому, в соответствии с описанными здесь вариантами осуществления изобретения, способы сбора информации о сцеплении генов могут быть осуществлены методами сцепления генов в близких положениях для определения пространственной взаимосвязи смежных генов; методами сцепления генов в положениях средней дальности друг от друга для определения пространственной взаимосвязи отдельных генов; методами сцепления генов, расположенных далеко друг от друга для определения пространственной взаимосвязи генов на далеких расстояниях. Эти методы облегчают оценку точности и качества сборки или картирования последовательностей ДНК и могут быть применены в комбинации с любым методом секвенирования, описанным выше.
Получение информации о сцеплении генов включвает сбор информации о родственных геномных ориджинах или координатах ридов отдельных последовательностей, так как они находятся в пространственной взаимосвязи между двумя или более фрагментами ДНК, от которых происходят риды отдельных последовательностей. В некоторых вариантах осуществления изобретения, получение информации о сцеплении генов включвает сбор информации о последовательностях неперекрывающихся ридов.
В некоторых вариантах осуществления изобретения, информация о сцеплении последовательностей нуклеиновой кислоти-мишени представляет собой информацию о гаплотипе. В некоторых вариантах осуществления изобретения, информация о сцеплении последовательности нуклеиновой кислоты-мишени представляет собой информацию о геномных вариантах.
Используемый здесь термин «сохранение сцепления нуклеиновой кислоти-мишени» при фрагментировании нуклеиновой кислоты означает сохранение порядка расположения фрагментов нуклеиновой кислоты в одной и той же нуклеиновой кислоте-мишени.
Используемый здесь термин «по меньшей мере часть» и/или его грамматические эквиваленты может означать любую часть от всего количества. Так, например, «по меньшей мере часть» может означать по меньшей мере приблизительно 1%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9%, 10%, 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95%, 99%, 99,9% или 100% от всего количества.
Используемый здесь термин «приблизительно» означает ±10%.
Используемый здесь термин «секвенирующий рид» и/или его грамматические эквиваленты может относиться к повторяющимся физическим или химическим стадиям, проводимым для получения сигналов, указывающих на порядок расположения мономеров в полимере. Сигналы могут указывать на порядок расположения мономеров при разрешении одного мономера или при более низком разрешении. В конкретных вариантах осуществления изобретения, стадии могут быть инициированы на нуклеиновой кислоте-мишени и осуществлены для получения сигналов, указывающих на порядок расположения оснований в нуклеиновой кислоте-мишени. Такой способ может быть осуществлен до его типичного завершения, обычно определяемого точкой, при которой сигналы больше не могут распознавать основания мишени с достаточной степенью достоверности. При необходимости, завершение процедуры может быть осуществлено раньше, например, после получения нужного количества информации о последовательности. Секвенирование рида может быть осуществлено на одной молекуле нуклеиновой кислоты-мишени или одновременно на группе молекул нуклеиновой кислоты-мишени, имеющих одну и ту же последовательность, или одновременно на группе нуклеиновых кислот-мишеней, имеющих различные последовательности. В некоторых вариантах осуществления изобретения, секвенирование рида прекращают после того, когда уже не могут быть получены сигналы от одной или более молекул нуклеиновой кислоты-мишени, от которых исходит этот сигнал. Так, например, секвенирование рида может быть инициировано для одной или более молекул нуклеиновой кислоты-мишени, присутствующих на твердофазном субстрате, и завершено после удаления одной или более молекул нуклеиновой кислоты-мишени из субстрата. Секвенирование может быть завершено посредством какого-либо иного способа прекращения детектирования нуклеиновых кислот-мишеней, присутствующих на субстрате в начале раунда секвенирования. Репрезентативные методы секвенирования описаны в патенте США No. 9029103, который во всей своей полноте вводятся в настоящее описание посредством ссылки.
Используемый здесь термин «представление секвенирования» и/или его грамматические эквиваленты могут означать информацию о порядке и типе мономерных единиц в полимере. Так, например, такая информация может указывать на порядок и тип нуклеотидов в нуклеиновой кислоте. Информация может быть представлена в любом формате, включая, но не ограничиваясь ими, рисунок, изображение, электронный носитель, ряд символов, ряд чисел, ряд букв, цветовую гамму и т.п. Информация может быть получена при разрешении одного мономера или при более низком разрешении. Репрезентативным полимером является нуклеиновая кислота, такая как ДНК или РНК, имеющая нуклеотидные единицы. Ряд букв «A», «T», «G» и «C» означает хорошо известное представление последовательности ДНК, которое может быть скорректировано при разрешении одного нуклеотида в фактической последовательности молекулы ДНК. Другими репрезентативными полимерами являются белки, имеющие аминокислотные звенья, и полисахариды, имеющие сахаридные звенья.
Твердый носитель
Во всем описании настоящей заявки, термины «твердый носитель» и «твердая поверхность» являются синонимами. В некоторых вариантах осуществления изобретения, твердый носитель или его поверхность не являются плоскими, то есть, представляют собой внутреннюю или внешнюю поверхность пробирки или сосуда. В некоторых вариантах осуществления изобретения, твердый носитель включает микросферы или сферы. Используемый здесь термин «микросферы» или «сферы» или «частицы» или его грамматические эквиваленты означают небольшие дискретные частицы. Подходящими материалами для сфер являются, но не ограничиваются ими, пластик, керамические изделия, стекло, полистирол, метилстирол, акриловые полимеры, парамагнитные материалы, соль тория, графит, диоксид титана, латекс или перекрестно-связанные декстраны, такие как сефароза, целлюлоза, найлон, перекрестно-связанные мицеллы и тефлон, а также любые другие материалы, которые могут быть применены для получения твердых носителей. Ценным руководством является «Руководство по детектированию микросфер» («Microsphere Detection Guide», Bangs Laboratories, Fishers Ind.). В некоторых вариантах осуществления изобретения, микросферы представляют собой магнитные микросферы или сферы. В некоторых вариантах осуществления изобретения, сферы могут иметь цветовые коды. Так, например, могут быть использованы микросферы MicroPlex®, поставляемые Luminex, Austin, TX.
Сферы необязательно должны иметь сферическую форму, например, могут быть использованы частицы неправильной формы. Альтернативно или дополнительно, сферы могут быть пористыми. Сферы имеют размеры от нанометров, то есть, приблизительно 10 нм, до миллиметров в диаметре, то есть, 1 мм, причем, предпочтительными являются сферы размером приблизительно от 0,2 микрона до 200 микрон, а особенно предпочтительно, приблизительно от 0,5 до 6 микрон, хотя в некоторых вариантах осуществления изобретения могут быть использованы сферы меньшего или большего размера. В некоторых вариантах осуществления изобретения, диаметр сфер может составлять приблизительно 0,1, 0,2, 0,3, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9, 1, 1,5, 2, 2,5, 2,8, 3, 3,5, 4, 4,5, 5, 5,5, 6, 6,5, 7, 7,5, 8, 8,5, 9, 9,5, 10, 10,5, 15, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 80, 85, 90, 95, 100, 150 или 200 мкм.
Транспозомы
«Транспозома» включает интергрирующий фермент, такой как интеграза или транспозаза и нуклеиновую кислоту, содержащую сайт распознавания интеграции, такой как сайт распознавания транспозазы. В описанных здесь вариантах, транспозаза может образовывать функциональный комплекс с сайтом распознавания транспозазы, способным катализировать реакции транспозиции. Транспозаза может связываться с сайтом распознавания транспозазы и встраивать сайт распознавания транспозазы в нуклеиновую кислоту-мишень по механизму, иногда называемому «тагментацией». В некоторых таких процедурах встраивания, одна цепь сайта распознавания транспозазы может быть перенесена в нуклеиновую кислоту-мишень. В одном из примеров, транспозома содержит димерную транспозазу, включающую две субъединицы и две несмежных транспозонных последовательности. В лругом примере, транспозома содержит транспозазу, включающую димерную транспозазу, содержащую две субъединицы и смежную транспозонную последовательность.
Некоторые варианты изобретения могут включать применение гиперактивной транспозазы Tn5 и сайта распознавания транспозазы Tn5-типа (Goryshin and Reznikoff, J. Biol. Chem., 273:7367 (1998)), или транспозазы MuA и сайта распознавания транспозазы Mu, содержащего концевые последовательности R1 и R2 (Mizuuchi, K., Cell, 35: 785, 1983; Savilahti, H, et al., EMBO J., 14: 4893, 1995). Репрезентативный сайт распознавания транспозазы, который образует комплекс с гиперактивной транспозазой Tn5 (например, EZ-Tn5™ Transposase, Epicentre Biotechnologies, Madison, Wisconsin), включает нижеследующие перенесенные цепи из 19 оснований (иногда обозначаемые «M» или «ME») и неперенесенные цепи: 5′ AGATGTGTATAAGAGACAG 3′ и 5′ CTGTCT CTTATACACATCT 3′, соответственно. Последовательности ME могут быть также использованы после их опитимизации специалистом.
Другими примерами систем транспозиции, которые могут быть применены в некоторых вариантах описанных здесь композиций и способов, являются Tn552 Staphylococcus aureus (Colegio et al., J. Bacteriol., 183: 2384-8, 2001; Kirby C et al., Mol. Microbiol., 43: 173-86, 2002), Ty1 (Devine & Boeke, Nucleic Acids Res., 22: 3765-72, 1994 и публикация Международной заявки WO 95/23875), транспозон Tn7 (Craig, N L, Science. 271: 1512, 1996; Craig, N L, Review in: Curr Top Microbiol Immunol., 204:27-48, 1996), Tn/O и IS10 (Kleckner N, et al., Curr Top Microbiol Immunol., 204:49-82, 1996), транспозаза Mariner (Lampe D J, et al., EMBO J., 15: 5470-9, 1996), Tc1 (Plasterk R H, Curr. Topics Microbiol. Immunol., 204: 125-43, 1996), элемент P (Gloor, G. B, Methods Mol. Biol., 260: 97-114, 2004), Tn3 (Ichikawa & Ohtsubo, J. Biol. Chem. 265:18829-32, 1990), бактериальные встраиваемые последовательности (Ohtsubo & Sekine, Curr. Top. Microbiol. Immunol. 204: 1-26, 1996), ретровирусы (Brown, et al., Proc. Natl. Acad. Sci. USA, 86:2525-9, 1989) и ретротранспозон дрожжей (Boeke & Corces, Annu Rev Microbiol. 43:403-34, 1989). Другими примерами являются IS5, Tn10, Tn903, IS911, Sleeping Beauty, SPIN, hAT, PiggyBac, Hermes, TcBuster, AeBuster1, Tol2 и сконструированные варианты ферментов семейства транспозаз (Zhang et al., (2009) PLoS Genet. 5:e1000689. Epub 2009 Oct 16; Wilson C. et al (2007) J. Microbiol. Methods 71:332-5).
Другими примерами интеграз, которые могут быть применены в описанных здесь способах и композициях, являются ретровирусные интегразы и последовательности распознавания таких ретровирусных интеграз, например, интеграз, происходящих от ВИЧ-1, ВИЧ-2, SIV, PFV-1, RSV.
Штрих-коды
Обычно, штрих-код может влючать одну или более нуклеотидных последовательностей, которые могут быть использованы для идентификации одной или более конкретных нуклеиновых кислот. Штрих-код может представлять собой искусственноую последовательность, либо он может представлять собой природную последовательность, образованную в процессе транспозиции, такую как идентичные фланкирующие геномные последовательности ДНК (г-коды) на конце ранее образованных фрагментов ДНК, находящихся в юкста-положении. В некоторых вариантах осуществления изобретения, штрих-код представляют собой искусственные последовательности, которые отсутствуют в последовательности нуклеиновой кислоты-мишени и могут быть использованы для идентификации одной или более последовательностей нуклеиновой кислоты-мишени.
Штрих-код может содержать по меньшей мере приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20 или более смежных нуклеотидов. В некоторых вариантах осуществления изобретения, штрих-код содержит по меньшей мере приблизительно 10, 20, 30, 40, 50, 60, 70 80, 90, 100 или более смежных нуклеотидов. В некоторых вариантах осуществления изобретения, по меньшей мере часть штрих-кодов в группе нуклеиновых кислот, имеющих эти штрих-коды, отличаются друг от друга. В некоторых вариантах осуществления изобретения, по меньшей мере приблизительно 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95%, 99% штрих-кодов являются различными. В других таких вариантах, все штрих-коды являются различными. Разнообразие различных штрих-кодов в группе нуклеиновых кислот, имеющих эти штрих-коды, может быть создано рандомизированным или нерандомизированным методом.
В некоторых вариантах осуществления изобретения, транспозонная последовательность включает по меньшей мере один штрих-код. В некоторых вариантах осуществления изобретения, транспозомы включают две несцепленных транспозонных последовательности, где первая транспозонная последовательность содержит первый штрих-код, а вторая транспозонная последовательность содержит второй штрих-код. В некоторых вариантах осуществления изобретения, транспозонная последовательность включает штрих-код, содержащий первую последовательность штрих-кода и вторую последовательность штрих-кода. В некоторых вышеуказанных вариантах, первая последовательность штрих-кода может быть идентифицирована или сконструирована так, чтобы она спаривалась со второй последовательностью штрих-кода. Так, например, известная первая последовательность штрих-кода может быть присоединена ко второй известной последовательности штрих-кода с использованием эталонной таблицы, включающей множество первых и вторых последовательностей штрих-кодов, которые, как известно, будут спариваться друг с другом.
В другом примере, первая последовательность штрих-кода может включать такую же последовательность штрих-кода, как и вторая последовательность штрих-кода. В другом примере, первая последовательность штрих-кода может включать обратный комплемент второй последовательности штрих-кода. В некоторых вариантах осуществления изобретения, первая последовательность штрих-кода и вторая последовательность штрих-кода являются различными. Первая и вторая последовательности штрих-кода могут содержать двоичный код.
В некоторых вариантах описанных здесь композиций и способов, штрих-коды используют для получения матричных нуклеиновых кислот. Совершенно очевидно, что очень большое число доступных штрих-кодов позволяет присвоить каждой матричной молекуле нуклеиновой кислоты уникальный идентификационный номер. Уникальная идентификация каждой молекулы в смеси матричных нуклеиновых кислот может быть использована в различных целях. Так, например, уникально идентифицированные молекулы могут быть применены для идентификация отдельных молекул нуклеиновой кислоты; образцов, имеющих множество хромосом; геномов; клеток; типов клеток; клеточных патологий, а в частности, например, для секвенирования гаплотипов, для дифференциации родительских аллелей, для секвенирования метагенома и для секвенирования генома в образце.
Репрезентативными последовательностями штрих-кодов являются, но не ограничиваются ими, TATAGCCT, ATAGAGGC, CCTATCCT, GGCTCTGA, AGGCGAAG, TAATCTTA, CAGGACGT и GTACTGAC.
Сайты праймеров.
В некоторых вариантах осуществления изобретения, транспозонная последовательность может включать «секвенирующий адаптер» или «сайт секвенирующего адаптера», то есть, можно сказать, что он содержит область, включающую один или более сайтов, которые могут гибридизоваться с праймером. В некоторых вариантах осуществления изобретения, транспозонная последовательность может включать по меньшей мере первый сайт праймера, используемый для амплификации, секвенирования и т.п. Репрезентативными последовательностями сайтов связывания с последовательностью являются, но не ограничиваются ими, AATGATACGGCGACCACCGAGATCTACAC (последовательность P5) и CAAGCAGAAGACGGCATACGAGAT (последовательность P7).
Нуклеиновые кислоты-мишени
Нуклеиновой кислотой-мишенью может быть любая представляющая интерес нуклеиновая кислота. Нуклеиновыми кислотами-мишенями могут быть ДНК, РНК, пептид-содержащая нуклеиновая кислота, морфолино-нуклеиновая кислота, блокированная нуклеиновая кислота, гликолевая нуклеиновая кислота, нуклеиновая кислота, содержащая треозу, смешанные образцы нуклеиновых кислот, полиплоидная ДНК (то есть, ДНК растения), а также их смеси и гибриды. В предпочтительном варианте осуществления изобретения, в качестве нуклеиновой кислоты-мишени используются гемномная ДНК или ее амплифицированные копии. В другом предпочтительном варианте осуществления изобретения используются кДНК, митохондриальная ДНК или ДНК хлоропластов. В некоторых вариантах осуществления изобретения, нуклеиновой кислотой-мишенью является мРНК.
В некоторых вариантах осуществления изобретения, нуклеиновая кислота-мишень происходит от одной клетки или из фракций одной клетки. В некоторых вариантах осуществления изобретения, нуклеиновая кислота-мишень происходит от одной органеллы. Репрезентативными моноорганеллами являются, но не ограничиваются ими, одно ядро, одна митохондрия и одна рибосома. В некоторых вариантах осуществления изобретения, нуклеиновую кислоту-мишень выделяют из залитого в парафин образца, фиксированного формалином (FFPE). В некоторых вариантах осуществления изобретения, нуклеиновой кислотой-мишенью является перекрестно-связанная нуклеиновая кислота. В некоторых вариантах осуществления изобретения, нуклеиновая кислота-мишень перекрестно связана с белком. В некоторых вариантах осуществления изобретения, нуклеиновой кислотой-мишенью является перекрестно-связанная ДНК. В некоторых вариантах осуществления изобретения, нуклеиновой кислотой-мишенью является ДНК, защищенная гистоном. В некоторых вариантах осуществления изобретения, гистоны удаляют из нуклеиновой кислоты-мишени. В некоторых вариантах осуществления изобретения, нуклеиновую кислоту-мишень выделяют из нуклеосом. В некоторых вариантах осуществления изобретения, нуклеиновую кислоту-мишень выделяют из нуклеосом, из которых были удалены ядерные белки.
Нуклеиновая кислота-мишень может содержать любую нуклеотидную последовательность. В некоторых вариантах осуществления изобретения, нуклеиновая кислота-мишень содержит гомополимерные последовательности. Нуклеиновая кислота-мишень может включать повторяющиеся последовательности. Повторяющиеся последовательности могут иметь любую длину, включая, например, 2, 5, 10, 20, 30, 40, 50, 100, 250, 500 или 1000 нуклеотидов или более. Повторяющиеся последовательности могут повторяться либо непрерывно, либо с перерывами любое количество раз, например, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15 или 20 раз или более.
В некоторых описанных здесь вариантах осуществления изобретения может быть использована одна нуклеиновая кислота-мишень. В других вариантах осуществления изобретения может быть использовано множество нуклеиновых кислот-мишеней. В таких вариантах осуществления изобретения, множество нуклеиновых кислот-мишеней млжет включать множество одних и тех же нуклеиновых кислот-мишеней, множество различных нуклеиновых кислот-мишеней, где некоторые нуклеиновые кислоты-мишени являются одинаковыми, или множество нуклеиновых кислот-мишеней, где все нуклеиновые кислоты-мишени являются различными. Варианты, в которых используется множество нуклеиновых кислот-мишеней, могут быть осуществлены в мультиплексном формате, так, чтобы реагенты могли доставляться одновременно к нуклеиновым кислотам-мишеням, например, в одну или более камер или на поверхность массива. В некоторых вариантах осуществления изобретения, множество нуклеиновых кислот-мишеней может включать, по существу, весь геном конкретного организма. Множество нуклеиновых кислот-мишеней может включать по меньшей мере часть генома конкретного организма, например, по меньшей мере приблизительно 1%, 5%, 10%, 25%, 50%, 75%, 80%, 85%, 90%, 95% или 99% генома. В конкретных вариантах осуществления изобретения, эта часть может иметь верхний предел, составляющий максимум приблизительно 1%, 5%, 10%, 25%, 50%, 75%, 80%, 85%, 90%, 95%, или 99% генома.
Нуклеиновые кислоты-мишени могут быть получены из любого источника. Так, например, нуклеиновые кислоты-мишени могут быть получены из молекул нуклеиновой кислоты, выделенных из одного организма, или из групп молекул нуклеиновой кислоты, выделенных из природных источников, которые включают один или более организмов. Источниками молекул нуклеиновой кислоты являются, но не ограничиваются ими, органеллы, клетки, ткани, органы или организмы. Клетками, которые могут служить в качестве источников молекул нуклеиновой кислоты-мишени, могут быть прокариотические клетки (бактериальные клетки, например, клетки бактерий рода Escherichia, Bacillus, Serratia, Salmonella, Staphylococcus, Streptococcus, Clostridium, Chlamydia, Neisseria, Treponema, Mycoplasma, Borrelia, Legionella, Pseudomonas, Mycobacterium, Helicobacter, Erwinia, Agrobacterium, Rhizobium и Streptomyces); археоциты, такие как кренархеоциты, наноархеоциты или эуриархеоциты; или эукариотические клетки, такие как клетки грибов (например, дрожжей), растений, простейших и других паразитов, а также клетки жвотных (включая насекомых (например, Drosophila spp.), нематод (например, Caenorhabditis elegans) и млекопитающих (например, крыс, мышей, обезьян, приматов, не являющихся человеком, и человека). Нуклеиновые кислоты-мишени и матричные нуклеиновые кислоты могут быть обогащены некоторым представляющими интерес последовательностями с применением различных методов, хорошо известных специалистам. Примеры таких методов описаны в публикации Международной заявки No. WO/2012/108864, которая во всей своей полноте вводится в настоящее описание посредством ссылки. В некоторых вариантах осуществления изобретения, нуклеиновые кислоты могут быть дополнительно обогащены в процессе получения матричных библиотек. Так, например, нуклеиновые кислоты могут быть обогащены некоторыми последовательностями до встраивания транспозом, после встраивания транспозом и/или после амплификации нуклеиновых кислот.
Кроме того, в некоторых вариантах осуществления изобретения, нуклеиновые кислоты-мишени и/или матричные нуклеиновые кислоты могут быть подвергнуты высокой степени очистки, например, нуклеиновые кислоты могут по меньшей мере приблизительно на 70%, 80%, 90%, 95%, 96%, 97%, 98%, 99% или 100% не содержать примесей, имеющихся до проведения описанных здесь способов. В некоторых вариантах осуществления изобретения, предпочтительно, применять способы, которые, как известно специалистам, обеспечивают сохранение качества и размера нуклеиновой кислоты-мишени, например, выделение и/или прямая транспозиция ДНК-мишени могут быть осуществлены в агарозных слоях. Транспозиция может быть также осуществлена непосредственно в клетках, в популяции клеток, в лизатах и в неочищенной ДНК.
В некоторых вариантах осуществления изобретения, нуклеиновая кислота-мишень может быть получена из биологического образца или образца, взятого у пациента. Используемый здесь термин «биологический образец» или «образец, взятый у пациента» включает образцы, такие как ткани и физиологические жидкости. Термин «физиологические жидкости» может включать, но не ограничивается ими, кровь, сыворотку, плазму, слюну, цереброспинальную жидкость, плевральные выпоты, слезы, грудное молоко, лимфу, мокроту, мочу, амниотическую жидкость и сперму. Образец может включать физиологическую жидкость, которая является «неклеточной». Термин «неклеточная физиологическая жидкость» включает менее, чем приблизительно 1% (масс./масс.) от всего клеточного материала. Примерами неклеточных физиологических жидкостей являются плазма или сыворотка. Образцом может быть образец природного или синтетического происхождения (то есть, клеточный образец, превращенный во внеклеточный).
В некоторых вариантах вышеописанных способов, нуклеиновая кислота-мишень может быть фрагментирована (например, путем обработки ультразвуком, гидролиза рестриктирующими ферментами или другими механическими способами) до ее обработки транспозомами.
Используемый здесь термин «плазма» означает неклеточную жидкость, присутствующую в крови. «Плазма» может быть выделена из крови путем удаления всех клеточных элементов крови методами, известными специалистам (например, путем центрифугировния, фильтрации и т.п.).
Используемые во всем описании артикли «a» или «an» означают «один или более», если это не оговорено особо.
Используемые здесь словосочетания «например», «так, например», «такой как», «включает», «включая» или их варианты не должны рассматриваться как ограничение и должны интерпретироваться как «не ограничивающие» или «без ограничений».
В нижеследующих примерах описаны иллюстративные варианты осуществления изобретения, которые не должны рассматриваться как ограничение объема описанного здесь изобретения.
Примеры
Пример 1. Выход ДНК-кластера, достигаемый с применением способа тагментации на сферах
Выход ДНК-кластера оценивали методом тагментации на сферах как показано на фигуре 3, и результаты такой оценки представлены в таблице на фигуре 4. В этом примере, 50, 250 и 1000 нг человеческой ДНК NA12878 тагментировали с использованием одной и той же партии сфер для тагментации (2,8 мкм-сферы). Вторую 50 нг-аликвоту ДНК NA12878 тагментировали с использованием второй партии сфер для тагментации (полный повтор; 2,8 мкм-сферы). Тагментированные образцы ДНК, связанные со сферами, подвергали ПЦР-амплификации и очищали. Аликвоту (5,4 мкл) каждого очищенного ПЦР-продукта (количественно не оцененного) подвергали 270-кратному разведению и получали маточные растворы образцов в концентрации приблизительно 50 пМ. Для каждого образца, 50 пМ маточного раствора разводили до 15, 19, 21 и 24 пМ. Разведенные образцы загружали на проточную кювету для получения и секвенирования кластера. Полученные данные показали, что при одном и том же разведении (~50 пМ), число кластеров составляло 100-114% для трех различных исходных уровней (то есть, 50, 250 и 1000 нг) при использовании одной и той же серии сфер. Число кластеров для 50 нг полного повтора (на другой партии сфер) составляло 81%. Другие разведения (15, 19, 21 и 24 пМ) давали одно и то же число кластеров в концентрации приблизительно 10%. Полученные данные показали, что сферы играют значительноую роль в регуляции выхода, и такой выход является воспроизводимым для различных исходных ДНК и для различных повторов.
Пример 2. Воспроизводимость способа тагментации на сферах
Воспроизводимость способа тагментации на сферах, проиллюстрированного на фигуре 3, показана на фигуре 5. В этом примере, для получения тагментированной ДНК с использованием 50 и 500 нг исходной ДНК NA12878 брали шесть различных препаратов индексированных сфер (помеченных цифрами от 1 до 6; 2,8 мкм-сферы), полученных при «одной и той же» плотности транспозом. Тагментированную ДНК подвергали ПЦР-амплификации и очищали. 12 очищенных ПЦР-продуктов объединяли с получением двух смесей (пула 1 и пула 2) из шести препаратов на двух дорожках HiSeq. Каждый пул включал 3-50 нг и 3-500 нг образцов на дорожку. Данные в таблице 500 представляют собой медианный размер вставки и средний размер вставки для каждого индексированного образца.
Пример 3. Размер вставки пула 1 и размер вставки пула 2
Размер вставки пула 1 и размер вставки пула 2 представлены на фигуре 6A (график 600) и представлены на фигуре 6В (график 650), соответственно, для индексированных образцов, проиллюстрированных на фигуре 5. Эти данные также показали, что размер вставок равномерно распределен по шести различным препаратам индексированных сфер. Тагментация на сферах представляет собой механизм регуляции размера вставок и выхода ДНК.
Пример 4. Воспроизводимость общего числа ридов.
Воспроизводимость общего числа ридов и процента выровненных ридов в эксперименте, описанном на фигуре 5, проиллюстрирована на фигуре 7 (гистограмма 700). При обеих исходных концентрациях (50 нг и 500 нг), общее число ридов является аналогичным для одного и того же препарата, полученного на индексированных сферах. Четыре из шести препаратов, полученных на индексированных сферах (индексы 1, 2, 3 и 6), имеют почти одинаковые выходы; при этом препараты на индексированных сферах 4 и 5 имеют некоторую степень вариабельности, которая может быть обусловлена присутствием индексной последовательности.
В одном из применений, способ тагментации на сферах может быть применен в анализе на обогащение экзома, где указанный анализ включает стадию тагментации, например, осуществляемую в соответствии с протоколом Illumina's Nextera® Rapid Capture Enrichment. В этом анализе на обогащение экзома (то есть, проводимом по протоколу Illumina's Nextera® Rapid Capture Enrichment), для фрагментирования геномной ДНК была проведена тагментация в растворе (Nextera). Затем для получения представляющих специфических генных фрагментов использовали геноспецифические праймеры. После этого осуществляли два цикла обогащения, а затем полученные фрагменты обогащали с помощью ПЦР и секвенировали.
Для оценки возможности применения способа тагментации на сферах в анализе на обогащение экзома, человеческую ДНК NA12878 тагментировали с использованием 25, 50, 100, 150, 200 и 500 нг исходной ДНК. Контрольную библиотеку (NA00536) получали из 50 нг исходной ДНК в соответствии со стандартным протоколом. Каждая исходная ДНК имеет свой индекс (уникальный идентификатор). В соответствии со стандартными методами и для гарантии получения достаточного количества фрагментов проводили десять циклов ПЦР с использованием активированной маточной смеси полимераз (EPM). В соответствии с протоколом, амплификацию проводили в течение 3 минут при 72°C, 30 секунд при 98°C, а затем 10 циклов по 10 секунд при 98°C, 30 секунд при 65°C и 1 минуту при 72°C. Затем образцы хранили при 10°C. После этого, образцы обрабатывали методом обогащения экзома и секвенировали.
Пример 5. Размер вставок в контрольной библиотеке и в тагментированной библиотеке на сферах в анализе на обогащение экзома
На фигурах 8A, 8B и 8C представлен график 800, на котором указан размер вставки в контрольной библиотеке, график 820, на котором указан размер вставки в тагментированной библиотеке на сферах, и данные, систематизированные в таблице 840, соответственно, для анализа на обогащение экзома. Эти данные показали, что тагментированные библиотеки на сферах имели более широкий диапазон размеров вставки по сравнению с контрольной библиотекой, но при этом, размер вставки почти не зависит от размера исходной ДНК в образцах.
Пример 6. Оценка качества последовательностей ридов
На фигурах 9A, 9B и 9C представлены: гистограмма 900, на которой представлены процент дупликаций, проходящих через фильтры (дупликаций PF); гистограмма 920 для PCT-выбранных оснований и гистограмма 940 для оснований, используемых в PCT на мишени, соответственно, в анализе на обогащение экзома, описанном на фигурах 8A, 8B и 8C. Как показано на фигуре 9A, процент дупликаций PF является показателем числа дуплицированных ридов на проточной кювете. В идеальном случае, это число будет низким (как в данном случае) и будет гарантировать, что все кластеры дадут данные, которые могут быть использованы для получения нужных результатов.
На фигуре 9B представлены PCT-отобранные основания, которые определяют число ридов, последовательности которых расположены в представляющем интерес сайте или поблизости от этого сайта, и которые должны быть обогащены при проведении процедуры обогащения. В идеальном случае, это число будет близким к единице, что будет указывать на успешное осуществление процедуры обогащения и на то, что риды, которые не должны быть обогащены, не пройдут такую процедуру.
На фигуре 9C представлены основания, используемые для PCT на мишени, которые определяют число ридов, имеющих фактическую последовательность по всем представляющим интерес конкретным основаниям в обогащенной области. В идеальном случае, все обогащенные риды должны иметь последовательность по всем представляющим интерес конкретным основаниям в обогащенном риде, но из-за рандомизированного характера тагментации и вариабельности длин вставок, могут быть обогащены риды, которые не были полностью секвенированы по всей представляющей интерес области.
Для оптимизации распределения размеров вставок были применены два метода. В одном из примеров, для удаления фрагментов, которые являются слишком мелкими или слишком крупными, может быть применен метод SPRI-очистки. SPRI-очистка представляет собой способ удаления фрагментов, размер которых выше или ниже нужного размера, посредством селективной преципитации ДНК исходя из размера и удерживания осажденной или неосажденной ДНК, если это необходимо (то есть, первую стадию проводят для осаждения только той ДНК, размер которой больше нужного размера, и удерживания более мелких растворимых фрагментов). Затем, более мелкие фрагменты осаждают, и в это время нежелательные очень мелкие фрагменты (которые еще присутствуют в растворе) удаляют и осажденную ДНК удерживают, промывают и повторно солюбилизируют с получением ДНК нужных размеров. В другом примере, пространство активных транспозом на поверхности сфер может быть использовано для регуляции распределения размеров вставок. Так, например, бреши на поверхности сфер могут быть заполнены неактивными транспозомами (например, транспозомами, содержащими неактивные транспозоны).
Был проведен анализ сцепления в способе тагментации на сферах. В таблице 3 указано число случаев, в которых 0, 1, 2 или 3 рида присутствуют 1000 п.о.-окнах с общим индексом. Были получены сферы с 9 различными индексированными транспозомами, и эти сферы были использованы для тагментации небольшого количества человеческих ДНК. Полученные риды выравнивали и анализировали на их число в 1000 п.о.- или 10 т.п.о.-окнах, имеющих один и тот же индекс. Некоторые риды в небольшом окне с общим индексом могут продуцироваться случайно, и предсказание вероятности продуцирования таких ридов проиллюстрировано в таблице 3 и в таблице 4 в строке «случайное продуцирование». Числа в строке «Сферы» означают фактическое число ридов с общим индексом в 1000 п.о.-окне (таблица 3) или 10 т.п.о.-окне (таблица 4). Как показано в таблице 3 и таблице 4, фактическое число обнаруженных одинаковых индексов в 1000 п.о.-окне или в 10 т.п.о.-окне значительно выше, чем это ожидается в случае случайного продуцирования. «0» окон означает, число случаев, в которых конкретное 1000 п.о.-окно не имело индексированных ридов, картированных в этом окне. Это число является наибольшим, поскольку было секвенировано лишь очень небольшое количество человеческих геномов, а большинтсво окон не имело ридов, выровненных по этим окнам. «1» означает число случаев, в которых только один рид был картирован по 1000 п.о. (или по 10 т.п.о.)-окну; «2» означает число случаев, в которых 2 рида имеют общий индекс в 1000 п.о. (или по 10 т.п.о.)-окне; и т.п. Эти данные позволяют предположить, что во всех 1400 случаях, один и тот же фрагмент ДНК (свыше 10 т.п.о.) был тагментирован на одной и той же сфере по меньшей мере от двух до 5 раз из всех приблизительно 15000 событий тагментации. Поскольку эти фрагменты имеют общий индекс, то маловероятно, но возможно, что эти фрагменты происходят от одной и той же сферы. Если фрагменты имеют один и тот же индекс, то маловероятно, что они образовались случайно, а скорее всего, они происходят от одной и той же сферы.
| Таблица 3. Число ридов в 1000 п.о.-окнах с общим индексом | ||||
| 0 | 1 | 2 | 3 | |
| Сфера | 25913666 | 15220 | 305 | 7 |
| Случайное продуцирование |
25913334 | 15855 | 9 | 0 |
В таблице 4 указано число ридов (до 5) в 10 т.п.о.-окнах с общим индексом.
| Таблица 4. Число ридов в 10 т.п.о.-окнах с общим индексом | ||||||
| 0 | 1 | 2 | 3 | 4 | 5 | |
| Сфера | 2578669 | 12683 | 1267 | 169 | 28 | 3 |
| Случайное продуцирование |
2577012 | 15742 | 64 | 1 | 0 | 0 |
Пример 7. Отделение свободных транспозом от CPT-ДНК
После транспозиции, реакционную смесь, содержащую CPT-ДНК и свободные транспозомы, подвергали колоночной хроматографии на сефакриле S-400 и эксклюзионной хроматографии на сефакриле S-200, и полученные данные были представлены на фигуре 22. CPT-ДНК обозначали как NCP ДНК.
Пример 8. Оптимизация плотность зондов для захвата на сферах
Плотности зондов A7 и B7 для захвата были оптимизированы на 1 мкм-сферах, и результаты такой оптимизации представлены на фигуре 25. Дорожки 1 (A7) и 3 (B7) имели более высокие плотности зондов, а дорожки 2 (A7) и 4 (B7) имели плотности зондов 10000-100000 на 1 мкм-сферу. Продукт лигирования зонда для захвата с молекулой-мишенью анализировали в агарозном геле. Зонды с плотностью приблизительно 10000-100000 на сферу давали более высокую эффективность лигирования, чем зонды с более высокой плотностью.
Пример 9. Тесты на возможность получения индексированных секвенирующих библиотек CPT-ДНК на сферах методом внутримолекулярной гибридизации.
Транспозомы были получены путем смешивания транспозонов, имеющих последовательности для захвата A7' и B7', которые являются комплементарными последовательностям для захвата A7 и B7 на сферах с гиперактивной транспозазой Tn5. Высокомолекулярную геномную ДНК смешивали с транспозомами, в результате чего получали CPT-ДНК. Отдельно были получены сферы с иммобилизованными олигонуклеотидами: P5-A7, P7-B7 или P5-A7+P7-B7, где P5 и P7 представляют собой последовательности, связывающиеся с праймерами, а A7 и B7 представляют собой последовательности для захвата, комплементарные последовательностям A7' и B7', соответственно. Сферы, содержащие только P5-A7, только P7-B7, P5-A7+P7-B7 или смесь сфер P5-A7 и P7-B7, обрабатывали CPT-ДНК, и к реакционной смеси добавляли лигазу для определения эффективности гибридизации иммобилизованных олигонуклеотидов с перенесенной ДНК. Результаты представлены на фигуре 26. Секвенирование библиотек осуществляли только в том случае, когда P5-A7 и P7-B7 были иммобилизованы вместе на одной сфере (дорожка 4), на что указывали полосы высокомолекулярного продукта на агарозном геле. Результаты указывали на высокую эффективность внутримолекулярной гибридизации и доказали целесообразность получения индексированных секвенирующих библиотек CPT-ДНК на сферах методом внутримолекулярной гибридизации.
Пример 10. Тесты на целесообразность клонального индексирования.
Было получено несколько серий транспозом. В одной серии, гиперактивную транспозазу Tn5 смешивали с транспозонными последовательностями Tnp1, имеющими 5'-биотин, в результате чего получали транспозому 1. В другой серии, Tnp2, имеющие уникальный индекс 2, смешивали с 5'-биотином, в результате чего получали транспозому 2. В другой серии, гиперактивную транспозазу Tn5 смешивали с транспозонными последовательностями Tnp3, имеющими 5'-биотин, в результате чего получали транспозому 3. В другой серии, Tnp4, имеющие уникальный индекс 4, смешивали с 5'-биотином, в результате чего получали транспозому 4. Каждую из транспозом 1 и 2 и транспозом 3 и 4 отдельно смешивали со стрептавидиновыми сферами, в результате чего получали серию сфер 1 и серию сфер 2. Затем две серии сфер смешивали и инкубировали с геномной ДНК и буфером для тагментации, что приводило к стимуляции тагментации геномной ДНК. После этого осуществляли ПЦР-амплификацию тагментированных последовательностй. Амплифицированную ДНК секвенировали для анализа на встраивание индексных последовательностей. Если тагментация органичивается сферами, то большинство фрагментов будут иметь индексы Tnp1/Tnp2 и Tnp3/Tnp4. В случае внутримолекулярной гибридизации, фрагменты могут иметь индексы Tnp1/Tnp4, Tnp2/Tnp3, Tnp1/Tnp3 и Tnp2/Tnp4. Результаты секвенирования после 5 и 10 циклов ПЦР представлены на фигуре 27. Контроль имеет все четыре транспозона, смешанные друг с другом и иммобилизованные на сферах. Результаты показали, что большинство последовательностей имеют индексы Tnp1/Tnp2 или Tnp3/Tnp4, что подтверждает целесообразность клонального индексирования. У контроля, каких-либо различий в индексах не наблюдалось.
Пример 11. Транспозиция индексированных клональных сфер за одну реакцию
Было получено 96 серий индексированных сфер с транспозомами. Отдельные индексированные транспозомы были получены путем смешивания транспозона, содержащего олигонуклеотид, включающий мозаичную концевую последовательность Tn5 (ME) у 5'-конца и индексную последовательность. Отдельные индексированные транспозомы иммобилизовали на сферах посредством взаимодействия «стрептавидин-биотин». Транспозомы, присутствующие на сферах, промывали, и все 96 отдельно индексированных транспозом на сферах объединяли. Олигонуклеотиды, комплементарные МЕ-последовательности и содержащие индексную последовательность, гибридизовали с иммобилизованным олигонуклеотидом, в результате чего получали транспозоны с уникальными индексами. 96 серий индексированных клональных сфер с транспозомами объединяли и инкубировали с высокомолекулярной (HMW) геномной ДНК в присутствии буфера для тагментации Nextera в одной пробирке.
Сферы промывали и транспозазу удаляли путем обработки реакционной смеси 0,1% ДСН. Тагментированную ДНК амплифицировали с индексированными праймерами и секвенировали в проточной кювете PE HiSeq v2 с использованием набора для получения кластера TrueSeq v3, после чего данные секвенирования анализировали.
Наблюдалось образование кластеров или островков ридов. График, на котором проиллюстрированы наименьшие расстояния между ридами для каждой последовательности, указывал, в основном, на присутствие главных пиков, то есть, одного пика в присутствии кластера (проксимального пика) и другого пика между кластерами (дистального пика). Этот метод и его результаты схематически представлены на фигурах 30 и 31. Размеры островков составляют приблизительно 3-10 т.п.о. Процент охватываемых оснований составляет приблизительно 5-10%. Размеры вставок геномной ДНК составляют приблизительно 200-300 оснований.
Пример 12. Размеры библиотек для транспозом, присутствующих на сферах
Сначала транспозомы собирали в растворе путем смешивания первого олигонуклеотида, имеющего последовательность ME', второго олигонуклеотида, имеющего последовательность ME-штрих-код-P5/P7, и транспозазы Tn5. В первой стадии, первый олигонуклеотид, имеющий последовательность ME', биотинилировали у 3'-конца. Во второй стадии, олигонуклеотид, имеющий последовательность ME-штрих-код-P5/P7, биотинилировали у 5'-конца. К каждой из полученных транспозом в различных концентрациях (10 нМ, 50 нМ и 200 нМ) добавляли серии стрептавидиновых сфер так, чтобы транспозомы были иммобилизованы на этих стрептавидиновых сферах. Сферы промывали и добавляли высокомолекулярную геномную ДНК, а затем осуществляли тагментацию. В некоторых случаях, тагментированную ДНК обрабатывали 0,1% ДСН, а в других случаях, тагментированную ДНК не обрабатывали. Тагментированную ДНК подвергали ПЦР-амплификации за 5-8 циклов и секвенировали. Эта процедура схематически представлена на фигуре 32.
Как показано на фигуре 33, ДСН-обработка повышала эффективность амплификации и качество секвенирования. Олигонуклеотиды с 3'-биотином имели большие размеры библиотеки для транспозом.
На фигуре 34 проиллюстрировано влияние плотности поверхности транспозомы на размер вставки. Транспозомы с 5'-биотином имели меньшие размеры библиотеки и давали большее число побочных продуктов аутоинсерции.
Пример 13. Титрование исходной ДНК
Различные количества высокомолекулярной ДНК-мишени добавляли к клонально индексированным сферам с плотностью 50 мМ Tn5:транспозон, и инкубировали в течение 15 или 60 минут при 37°C или в течение 60 минут при комнатной температуре. Транспозомы состояли из олигонуклеотидов с 3'-биотином. Затем осуществляли тагментацию и реакционную смесь обрабатывали 0,1% ДСН и подвергали ПЦР-амплификации. Амплифицированную ДНК секвенировали. На фигуре 35 проиллюстрировано влияние исходной ДНК на распределение по размеру. Реакции с 10 пг исходной ДНК давали наименьший сигнал. Характер распределения по размеру был аналогичным для исходных ДНК в концентрации 20, 40 и 200 пг.
Пример 14. Размер и распределение островков, оцениваемые методами, осуществляемыми в растворе и на сферах
Было проведено сравнение размера и распределения островков, оцениваемых методами, осуществляемыми в растворе и на сферах. В методе, осуществляемом в растворе, 96 транспозом, каждая из которых имела транспозоны с уникальным индексом, подвергали сборке в 96-луночном планшете. Затем добавляли высокомолекулярную геномную ДНК и осуществляли реакцию тагментации. Реакционный продукт обрабатывали 0,1% ДСН и подвергали ПЦР-амплификации. Амплифицированные продукты секвенировали.
В методе, осуществляемом на сферах, 96 транспозом, каждая из которых имела транспозоны с уникальным индексом, подвергали сборке в 96-луночном планшете. Олигонуклеотиды состояли из 3'-концевого биотина. В каждый 96-луночный планшет добавляли стрептавидиновые сферы и смесь инкубировали так, чтобы транспозомы были иммобилизованы на стрептавидиновых сферах. Сферы отдельно промывали и собирали, а затем добавляли высокомолекулярную геномную ДНК и осуществляли реакцию тагментации в одном реакционном сосуде (в одном сосуде). Реакционный продукт обрабатывали 0,1% ДСН и подвергали ПЦР-амплификации. Амплифицированные продукты секвенировали.
В случае негативного контроля, все 96 транспозонных последовательностей, каждая из которых имела уникальный индекс, смешивали. Олигонуклеотиды состояли из 3'-концевого биотина. Транспозомы получали из отдельно смешанных индексированных транспозонов. Затем к смеси добавляли стрептавидиновые сферы. После этого добавляли высокомолекулярную геномную ДНК и осуществляли реакцию тагментации. Реакционный продукт обрабатывали 0,1% ДСН и подвергали ПЦР-амплификации. Амплифицированные продукты секвенировали.
Была построена кривая зависимости числа ридов внутри островков от размера островков. Результаты, представленные на фигуре 36, показали, что островки (проксимальные риды), наблюдаемые при использовании клональных индексированных сфер в одном сосуде, были аналогичны островкам, наблюдаемым при проведении метода в растворе. При смешивании индексированных транспозонов до образования транспозомы, островки (проксимальные риды) не наблюдались. Смешивание транспозонов до образования транспозомы приводило к образованию сфер с различными индексами/транспозомами на сферу, то есть, неклональных сфер.
Пример 15. Структурный анализ варианта с помощью CPT-seq
Детектирование гетерозиготной делеции в 60 т.п.о.
Данные секвенирования были представлены в виде файлов fastq и подвергнуты разделению для создания отдельного файла fastq для каждого штрих-кода. Файлы fastq, созданные после секвенирования CPT, подвергали разделению в соответствии с их индексами и сопоставляли путем выравнивания с эталонным геномом с последующим удалением дубликатов. Хромосомы сканировали по окну 5 т.п.о./1 т.п.о., в котором регистрировали число индексов, указывающих на то, что любые риды находятся в окне сканирования. С точки зрения статистики, для гетерозиготной области делеции, по сравнению с ее соседними областями, для создания библиотеки доступна только половина от всего количества ДНК, а поэтому число индексов должно составлять приблизительно половину, как их соседняя половина. Гетерозиготная делеция 60 т.п.о. в хромосоме 1 NA12878 была обнаружена после сканирования по окну 5 т.п.о. для 9216 индексированных данных секвенирования CPT и представлена на фиг. 47A и 47B.
Детектирование сцепления генов
Файлы fastq, созданные после секвенирования CPT, подвергали разделению в соответствии с их индексами и сопоставляли путем выравнивания с эталонным геномом с последующим удалением дубликатов. Хромосомы сканировали по 2 т.п.о.-окну. Каждое 2 т.п.о.-окно представляет собой вектор 36864, в котором каждый элемент регистрирует число ридов, имеющих уникальный индекс и присутствующих в этом 2 т.п.о.-окне. Для каждой пары 2 т.п.о.-окон (X,Y) по всему геному вычисляли взвешенный индекс Джаккарда. Этот индекс de facto указывает на расстояние между парами (X,Y) в образце. Эти индексы проиллюстрированы в виде тепловой карты, представленной на фиг. 48, где каждая экспериментальная точка представляет пару 2 т.п.о.-окон сканирования; левый верхний квадрат относится к паре X,Y области 1, нижний правый квадрат относится к паре X,Y области 2, а верхний правый квадрат относится к паре X,Y области пересечения областей 1 и 2. Сигнал сцепления генов представлен горизонтальной линией в середине этого графика.
Детектирование делеций
Файлы fastq, созданные после секвенирования CPT, подвергали разделению в соответствии с их индексами и сопоставляли путем выравнивания с эталонным геномом с последующим удалением дубликатов. Хромосомы сканировали по 2 т.п.о.-окну. Результаты детектирования генетических делеций представлены на фиг. 49.
Пример 16. Детектирование фазирования и метилирования
Оптимизация эффективности превращения под действием бисульфита
Превращение оценивали в области ME (области мозаичного элемента) и гДНК для сцепленных с индексом библиотек CPT-Seq на сферах. Систему превращения под действием бисульфита (Promega's MethylEdge Bisulfite Conversion system) оптимизировали для повышения эффективности.
| Условие | ДНК | Сферы | BSC-обработка |
| 1 | 10 нг | Нет | 1 час @ 60°C/0,3M NaOH |
| 2 | Да | 1 час @ 60°C/0,3M NaOH | |
| 3 | 1 час @ 60°C/1M NaOH | ||
| 4 | 1 час @ 65°C/0,3M NaOH |
ME-последовательности анализировали для определения эффективности обработки путем бисульфитного превращения, и результаты анализа представлены на фиг. 50. Бисульфитное превращение (BSC) для сцепленных с индексами библиотек, связанных со сферами, составляло 95%. Аналогичные ПЦР-выходы, наблюдаемые в условиях обработки дисульфитом > более жестких условиях обработки дисульфитом, не приводили к разложению библиотек, как показано на фиг. 51. При этом наблюдалось приблизительно 95%-ное BSC-превращение сцепленных с индексами библиотек на сферах. Переменными величинами, исследуемыми для оценки улучшения качества BSC (C®U), являются температура и концентрация NaOH (денатурация). Условия 60°C и 1M NaOH или°C и 0,3 M NaOH давали хороший результат.
После секвенирования BSC-превращенных CPT-seq-библиотек на сферах наблюдалась ожидаемая структура секвенирующего рида. Процентные соотношения оснований представлены на графике IVC на фиг. 52.
На фиг. 53 проиллюстрирован электрофорез в агарозном геле для сцепленных с индексами библиотек после проведения ПЦР и после бисульфитного превращения. При этом наблюдались библиотеки ожидаемого размера в пределах 200-500 п.о. Реакция, проводимая в отсутствии ДНК, не давала каких-либо сцепленных с индексами библиотек.
Пример 17. Целевое фазирование
Было проведено обогащение сцепленных с индексами библиотек CPT-seq во всем геноме. На фиг. 54 проиллюстрировано мечение с использованием биоанализатора для сцепленных с индексами библиотек CPT-seq во всем геноме перед обогащением без выбора по размеру. На фиг. 55 проиллюстрирован проводимый в агарозном геле анализ библиотек после обогащения.
Статистические методы обогащения для области HLA представлены ниже:
| Образец ID | C3 |
| Название образца: | HLA-зонды |
| Размер слоя: | 150 |
| Общая длина эталона мишени: | 5062748 |
| Общее число ридов, прошедших через фильтр (PF): | 2516 |
| Процент Q30: | 94,90% |
| Общее число выровненных ридов: | 2498 |
| Процент выровненных ридов: | 99,40% |
| Выровненные риды мишени: | 840 |
| Обогащение ридов: | 30,80% |
| Процент спаренных дуплицированных ридов: | 12,70% |
| Медианная длина фрагмента: | 195 |
На фигуре 56 представлены результаты целевого гаплотипирования области HLA в хромосоме. Слева проиллюстрировано обогащение библиотеки сцепленных с индексами ридов во всем геноме. Каждая небольшая гистограмма представляет короткую индексированную библиотеку. Кластерами индексированных библиотек являются «островки», то есть, области, которые были клонально индексированы на однгой сфере с одним и тем же индексом, и следовательно, эти риды являются проксимальными (типа «островков») на уровне генома. Обогащение (см. «Селективное обогащение нуклеиновых кислот» (Selective enrichment of nucleic acids) в заявке WO 2012108864 A1) библиотек в области мишени представлено справа. Риды обогащали для HLA-области. Кроме того, если риды были отобраны по индексам и выровнены с геномом, то их снова представляли в виде структуры «островков», что указывает на сохранение информации о целостности сцепленных с индексами ридов.
Пример 18. Замены индексов
Для оценки замен мозаичных концов (ME) транспозомных комплексов были получены сферы с различными индексами. После смешивания определяли замены индексов путем секвенирования библиотек, а затем регистрировали эти индексы для каждой библиотеки. Проценты «замененных» индексов были вычислены по формуле: (D4+D5+E3+E5+f4)/(сумма из всех 96) и представлены на фигуре 65.
Пример 19. Снижение размера вставки библиотеки путем получения более плотных групп транспозомных комплексов, связанных со стрептавидиновыми сферами
Стрептавидиновые магнитные сферы нагружали 1×, 6× и 12× концентрациями транспозомного комплекса TsTn5. Протокол секвенирования Epi-CPT осуществляли для сферы каждого типа. Конечный ПЦР-продукт загружали на биоанализатор Agilent BioAnalyzer для анализа, проиллюстрированного на фигуре. Фрагменты библиотек Epi-CPT seq были меньше и давали больший выход при загрузке большего количества TsTn5 на сферы.
Пример 20. Фрагментирование библиотеки ДНК в процессе превращения под действием бисульфата натрия
После превращения под действием бисульфита, ДНК разрушалась, что приводило к потере общих последовательностей (CS2), необходимых для ПЦР-амплификации. Библиотеки фрагментов ДНК CPTSeq и Epi-CPTSeq (Me-CPTSeq) анализировали на биоанализаторе. Из-за разрушения ДНК в процессе бисульфитного превращения, библиотека Epi-CPTSeq давала в 5 раз меньший выход и менее равномерное распределение по размеру по сравнению с библиотекой CPTSeq, как показано на фигуре 70.
Пример 21. TdT-опосредуемая реакция лигирования оцДНК
Была протестирована возможность восстановления концов ДНК посредством лигирования, опосредуемого концевой трансферазой (TdT). Вкратце, 5 пмоль матричной оцДНК инкубировали с TdT (10/50 ед.), с дуплексом «аттенюатор/адаптер» (0/15/25 пмоль) и с ДНК-лигазой (0/10 ед.) в течение 15 минут при 37°C. Продукты удлинения/лигирования ДНК анализировали на геле, содержащем TBE-мочевину, и полученные результаты были представлены на фигуре 71. Добавление всех реакционных компонентов приводило к почти полному лигированию молекулы адаптера (дорожки 5-8).
Возможность восстановления концов ДНК посредством лигирования, опосредуемого концевой трансферазой (TdT), была протестирована для связанной со сферой библиотеки, подвергнутой реакции превращения бисульфатом натрия, и результаты были представлены на фигуре 72. Вкратце, ДНК тагментировали на сферах (первые две дорожки), обрабатывали реагентами, входящими в набор для реакции превращения под действием бисульфата Promega's MethylEdge (дорожки 3 и 4) и подвергали процедуре «спасения» ДНК в соответствии с протоколом (дорожки 5 и 6). После реакции «спасения» наблюдалось явное увеличение выхода и размера библиотеки ДНК. При этом также наблюдалось увеличение количества аутоинсерционных транспозонов (SI), что указывало на эффективное лигирование молекулы адаптера.
Результаты анализа методом секвенирования метил-CPTSeq представлены на фигуре 73.
Claims (22)
1. Композиция для получения библиотеки, содержащая множество клонально индексированных твердых носителей,
где каждый из твердых носителей во множестве твердых носителей иммобилизует на себе множество олигонуклеотидов и
где каждый из множества олигонуклеотидов, иммобилизованных на указанном твердом носителе, содержит
комплементарную последовательность для захвата для иммобилизации на твердом носителе транспозомного комплекса, который ассоциирован c нуклеиновой кислотой-мишенью, следующей за смежно-связанной транспозицией нуклеиновой кислоты-мишени, где транспозомный комплекс содержит транспозон, связанный с транспозазой, и где сцепление фрагментов нуклеиновой кислоты- мишени, созданное транспозазами транспозомного комплекса, поддерживается транспозазами,
первую последовательность со штрих-кодом и сайт связывания с праймером,
где первая последовательность со штрих-кодом с каждого твердого носителя из множества твердых носителей отличается по существу от всех первых последовательностей со штрих-кодом с других твердых носителей во множестве твердых носителей,
где по существу все из олигонуклеотидов, мобилизованных на указанном твердом носителе, содержат одну и ту же комплементарную последовательность для захвата и
где по существу все из олигонуклеотидов, мобилизованных на указанном твердом носителе, содержат одну и ту же первую последовательность со штрих-кодом.
2. Композиция по п. 1, где комплементарная последовательность для захвата множества иммобилизованных олигонуклеотидов твердого носителя гибридизована с последовательностью адаптера транспозона транспозомного комплекса.
3. Композиция по п. 1, где транспозаза транспозомного комплекса представляет собой транспозазу Tn5.
4. Композиция по п. 1, где множество иммобилизованных олигонуклеотидов дополнительно включает вторую последовательность со штрих-кодом, необязательно имеющую сайт фрагментации, расположенный между указанной первой последовательностью со штрих-кодом и второй последовательностью со штрих-кодом.
5. Композиция по п. 4, где олигонуклеотиды дополнительно включают третью последовательность со штрих-кодом.
6. Композиция по п. 2, где последовательность адаптера транспозона содержит последовательность со штрих-кодом.
7. Композиция по п. 2, где последовательность адаптера транспозона содержит сайт связывания с праймером, необязательно где сайт связывания с праймером последовательности адаптера транспозона представляет собой сайт связывания с праймером P5 или P7.
8. Композиция по п. 1, где транспозомный комплекс связан с нуклеиновой кислотой-мишенью.
9. Композиция по п. 8, где транспозон содержит перенесенную цепь и неперенесенную цепь, где перенесенная цепь вставлена в нуклеиновую кислоту-мишень.
10. Композиция по п. 8, где нуклеиновая кислота-мишень содержит множество фрагментов и транспозомные комплексы связаны с множеством фрагментов.
11. Композиция по п. 10, где транспозон содержит перенесенную цепь и неперенесенную цепь, где перенесенная цепь вставлена в 5’ конец по меньшей мере одной цепи фрагментов с сохранением сцепления нуклеиновой кислоты-мишени с по меньшей мере одной их транспозазой.
12. Композиция по п. 3, где комплементарная последовательность для захвата олигонуклеотида лигирована с последовательностью адаптера транспозона.
13. Композиция по п. 1, где каждый из твердых носителей во множестве твердых носителей выбран из группы, состоящей из микросферы, сферы и частицы.
14. Композиция по п. 13, где каждый из твердых носителей во множестве твердых носителей представляет собой сферу, которая имеет диаметр 0,5-5 мкм.
15. Композиция по п. 1, где сайт связывания с праймером каждого иммобилизованного олигонуклеотида комплементарен секвенирующему паймеру, содержащему праймер P5 или P7.
Applications Claiming Priority (6)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201462065544P | 2014-10-17 | 2014-10-17 | |
| US62/065,544 | 2014-10-17 | ||
| US201562157396P | 2015-05-05 | 2015-05-05 | |
| US62/157,396 | 2015-05-05 | ||
| US201562242880P | 2015-10-16 | 2015-10-16 | |
| US62/242,880 | 2015-10-16 |
Related Parent Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| RU2017116989A Division RU2709655C2 (ru) | 2014-10-17 | 2015-10-16 | Транспозиция с сохранением сцепления генов |
Related Child Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| RU2020137454A Division RU2020137454A (ru) | 2014-10-17 | 2020-11-16 | Транспозиция с сохранением сцепления генов |
Publications (3)
| Publication Number | Publication Date |
|---|---|
| RU2019138705A RU2019138705A (ru) | 2020-01-27 |
| RU2019138705A3 RU2019138705A3 (ru) | 2020-05-22 |
| RU2736728C2 true RU2736728C2 (ru) | 2020-11-19 |
Family
ID=55747561
Family Applications (2)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| RU2019138705A RU2736728C2 (ru) | 2014-10-17 | 2015-10-16 | Транспозиция с сохранением сцепления генов |
| RU2017116989A RU2709655C2 (ru) | 2014-10-17 | 2015-10-16 | Транспозиция с сохранением сцепления генов |
Family Applications After (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| RU2017116989A RU2709655C2 (ru) | 2014-10-17 | 2015-10-16 | Транспозиция с сохранением сцепления генов |
Country Status (10)
| Country | Link |
|---|---|
| US (3) | US11873480B2 (ru) |
| JP (3) | JP6808617B2 (ru) |
| KR (2) | KR102643955B1 (ru) |
| AU (2) | AU2015331739B2 (ru) |
| BR (2) | BR122021026779B1 (ru) |
| CA (1) | CA2964799A1 (ru) |
| IL (3) | IL299976B1 (ru) |
| RU (2) | RU2736728C2 (ru) |
| SG (2) | SG11201703139VA (ru) |
| WO (1) | WO2016061517A2 (ru) |
Families Citing this family (146)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US8835358B2 (en) | 2009-12-15 | 2014-09-16 | Cellular Research, Inc. | Digital counting of individual molecules by stochastic attachment of diverse labels |
| US9074251B2 (en) | 2011-02-10 | 2015-07-07 | Illumina, Inc. | Linking sequence reads using paired code tags |
| WO2012106546A2 (en) | 2011-02-02 | 2012-08-09 | University Of Washington Through Its Center For Commercialization | Massively parallel continguity mapping |
| EP2820158B1 (en) | 2012-02-27 | 2018-01-10 | Cellular Research, Inc. | Compositions and kits for molecular counting |
| US10273541B2 (en) | 2012-08-14 | 2019-04-30 | 10X Genomics, Inc. | Methods and systems for processing polynucleotides |
| US11591637B2 (en) | 2012-08-14 | 2023-02-28 | 10X Genomics, Inc. | Compositions and methods for sample processing |
| US10752949B2 (en) | 2012-08-14 | 2020-08-25 | 10X Genomics, Inc. | Methods and systems for processing polynucleotides |
| US10400280B2 (en) | 2012-08-14 | 2019-09-03 | 10X Genomics, Inc. | Methods and systems for processing polynucleotides |
| US10323279B2 (en) | 2012-08-14 | 2019-06-18 | 10X Genomics, Inc. | Methods and systems for processing polynucleotides |
| JP6367196B2 (ja) | 2012-08-14 | 2018-08-01 | テンエックス・ジェノミクス・インコーポレイテッド | マイクロカプセル組成物および方法 |
| US9701998B2 (en) | 2012-12-14 | 2017-07-11 | 10X Genomics, Inc. | Methods and systems for processing polynucleotides |
| US9951386B2 (en) | 2014-06-26 | 2018-04-24 | 10X Genomics, Inc. | Methods and systems for processing polynucleotides |
| US10533221B2 (en) | 2012-12-14 | 2020-01-14 | 10X Genomics, Inc. | Methods and systems for processing polynucleotides |
| EP3567116A1 (en) | 2012-12-14 | 2019-11-13 | 10X Genomics, Inc. | Methods and systems for processing polynucleotides |
| US9644204B2 (en) | 2013-02-08 | 2017-05-09 | 10X Genomics, Inc. | Partitioning and processing of analytes and other species |
| WO2014142850A1 (en) | 2013-03-13 | 2014-09-18 | Illumina, Inc. | Methods and compositions for nucleic acid sequencing |
| US9328382B2 (en) | 2013-03-15 | 2016-05-03 | Complete Genomics, Inc. | Multiple tagging of individual long DNA fragments |
| GB2546833B (en) | 2013-08-28 | 2018-04-18 | Cellular Res Inc | Microwell for single cell analysis comprising single cell and single bead oligonucleotide capture labels |
| US10395758B2 (en) | 2013-08-30 | 2019-08-27 | 10X Genomics, Inc. | Sequencing methods |
| US9824068B2 (en) | 2013-12-16 | 2017-11-21 | 10X Genomics, Inc. | Methods and apparatus for sorting data |
| WO2015157567A1 (en) | 2014-04-10 | 2015-10-15 | 10X Genomics, Inc. | Fluidic devices, systems, and methods for encapsulating and partitioning reagents, and applications of same |
| KR102531677B1 (ko) | 2014-06-26 | 2023-05-10 | 10엑스 제노믹스, 인크. | 개별 세포 또는 세포 개체군으로부터 핵산을 분석하는 방법 |
| AU2015296029B2 (en) | 2014-08-01 | 2022-01-27 | Dovetail Genomics, Llc | Tagging nucleic acids for sequence assembly |
| AU2015339148B2 (en) | 2014-10-29 | 2022-03-10 | 10X Genomics, Inc. | Methods and compositions for targeted nucleic acid sequencing |
| US9975122B2 (en) | 2014-11-05 | 2018-05-22 | 10X Genomics, Inc. | Instrument systems for integrated sample processing |
| EP3244992B1 (en) | 2015-01-12 | 2023-03-08 | 10X Genomics, Inc. | Processes for barcoding nucleic acids |
| EP3259696A1 (en) | 2015-02-17 | 2017-12-27 | Dovetail Genomics LLC | Nucleic acid sequence assembly |
| US11274343B2 (en) | 2015-02-24 | 2022-03-15 | 10X Genomics, Inc. | Methods and compositions for targeted nucleic acid sequence coverage |
| EP4286516A3 (en) | 2015-02-24 | 2024-03-06 | 10X Genomics, Inc. | Partition processing methods and systems |
| CN107208158B (zh) | 2015-02-27 | 2022-01-28 | 贝克顿迪金森公司 | 空间上可寻址的分子条形编码 |
| US11807896B2 (en) | 2015-03-26 | 2023-11-07 | Dovetail Genomics, Llc | Physical linkage preservation in DNA storage |
| JP7508191B2 (ja) | 2015-03-30 | 2024-07-01 | ベクトン・ディキンソン・アンド・カンパニー | コンビナトリアルバーコーディングのための方法および組成物 |
| WO2016172373A1 (en) | 2015-04-23 | 2016-10-27 | Cellular Research, Inc. | Methods and compositions for whole transcriptome amplification |
| US11453875B2 (en) | 2015-05-28 | 2022-09-27 | Illumina Cambridge Limited | Surface-based tagmentation |
| WO2017004612A1 (en) | 2015-07-02 | 2017-01-05 | Arima Genomics, Inc. | Accurate molecular deconvolution of mixtures samples |
| WO2017034970A1 (en) * | 2015-08-21 | 2017-03-02 | The General Hospital Corporation | Combinatorial single molecule analysis of chromatin |
| EP3347465B1 (en) | 2015-09-11 | 2019-06-26 | Cellular Research, Inc. | Methods and compositions for nucleic acid library normalization |
| CA3002740A1 (en) | 2015-10-19 | 2017-04-27 | Dovetail Genomics, Llc | Methods for genome assembly, haplotype phasing, and target independent nucleic acid detection |
| US11371094B2 (en) | 2015-11-19 | 2022-06-28 | 10X Genomics, Inc. | Systems and methods for nucleic acid processing using degenerate nucleotides |
| DK3882357T3 (da) | 2015-12-04 | 2022-08-29 | 10X Genomics Inc | Fremgangsmåder og sammensætninger til analyse af nukleinsyrer |
| US10730030B2 (en) | 2016-01-08 | 2020-08-04 | Bio-Rad Laboratories, Inc. | Multiple beads per droplet resolution |
| WO2017123758A1 (en) * | 2016-01-12 | 2017-07-20 | Seqwell, Inc. | Compositions and methods for sequencing nucleic acids |
| WO2017138984A1 (en) | 2016-02-11 | 2017-08-17 | 10X Genomics, Inc. | Systems, methods, and media for de novo assembly of whole genome sequence data |
| CA3014911A1 (en) | 2016-02-23 | 2017-08-31 | Dovetail Genomics, Llc | Generation of phased read-sets for genome assembly and haplotype phasing |
| US20170283864A1 (en) | 2016-03-31 | 2017-10-05 | Agilent Technologies, Inc. | Use of transposase and y adapters to fragment and tag dna |
| CN110199019B (zh) | 2016-05-02 | 2024-09-10 | Encodia有限公司 | 采用核酸编码的大分子分析 |
| DK3455356T3 (da) * | 2016-05-13 | 2021-11-01 | Dovetail Genomics Llc | Genfinding af langtrækkende bindingsinformation fra konserverede prøver |
| WO2017197338A1 (en) | 2016-05-13 | 2017-11-16 | 10X Genomics, Inc. | Microfluidic systems and methods of use |
| US10894990B2 (en) | 2016-05-17 | 2021-01-19 | Shoreline Biome, Llc | High throughput method for identification and sequencing of unknown microbial and eukaryotic genomes from complex mixtures |
| US10301677B2 (en) | 2016-05-25 | 2019-05-28 | Cellular Research, Inc. | Normalization of nucleic acid libraries |
| US10202641B2 (en) | 2016-05-31 | 2019-02-12 | Cellular Research, Inc. | Error correction in amplification of samples |
| US10640763B2 (en) | 2016-05-31 | 2020-05-05 | Cellular Research, Inc. | Molecular indexing of internal sequences |
| WO2018013724A1 (en) * | 2016-07-12 | 2018-01-18 | Kapa Biosystems, Inc. | System and method for transposase-mediated amplicon sequencing |
| WO2018018008A1 (en) * | 2016-07-22 | 2018-01-25 | Oregon Health & Science University | Single cell whole genome libraries and combinatorial indexing methods of making thereof |
| AU2017311306A1 (en) * | 2016-08-10 | 2019-03-14 | President And Fellows Of Harvard College | Methods of de novo assembly of barcoded genomic DNA fragments |
| WO2018057779A1 (en) * | 2016-09-23 | 2018-03-29 | Jianbiao Zheng | Compositions of synthetic transposons and methods of use thereof |
| CA3034924A1 (en) | 2016-09-26 | 2018-03-29 | Cellular Research, Inc. | Measurement of protein expression using reagents with barcoded oligonucleotide sequences |
| CN118441026A (zh) | 2016-12-19 | 2024-08-06 | 生物辐射实验室股份有限公司 | 液滴加标的相邻性保留的标签化dna |
| US10550429B2 (en) | 2016-12-22 | 2020-02-04 | 10X Genomics, Inc. | Methods and systems for processing polynucleotides |
| US10815525B2 (en) | 2016-12-22 | 2020-10-27 | 10X Genomics, Inc. | Methods and systems for processing polynucleotides |
| GB201622219D0 (en) | 2016-12-23 | 2017-02-08 | Cs Genetics Ltd | Methods and reagents for molecular barcoding |
| WO2018140966A1 (en) | 2017-01-30 | 2018-08-02 | 10X Genomics, Inc. | Methods and systems for droplet-based single cell barcoding |
| WO2018144240A1 (en) | 2017-02-01 | 2018-08-09 | Cellular Research, Inc. | Selective amplification using blocking oligonucleotides |
| US10995333B2 (en) | 2017-02-06 | 2021-05-04 | 10X Genomics, Inc. | Systems and methods for nucleic acid preparation |
| WO2018156519A1 (en) * | 2017-02-21 | 2018-08-30 | Illumina Inc. | Tagmentation using immobilized transposomes with linkers |
| ES2973573T3 (es) | 2017-05-05 | 2024-06-20 | Scipio Bioscience | Métodos para atrapar e identificar con código de barras unidades biológicas discretas en hidrogel |
| US10844372B2 (en) | 2017-05-26 | 2020-11-24 | 10X Genomics, Inc. | Single cell analysis of transposase accessible chromatin |
| CN109526228B (zh) * | 2017-05-26 | 2022-11-25 | 10X基因组学有限公司 | 转座酶可接近性染色质的单细胞分析 |
| US10676779B2 (en) | 2017-06-05 | 2020-06-09 | Becton, Dickinson And Company | Sample indexing for single cells |
| SG11201911730XA (en) * | 2017-06-07 | 2020-01-30 | Univ Oregon Health & Science | Single cell whole genome libraries for methylation sequencing |
| WO2019060722A2 (en) | 2017-09-22 | 2019-03-28 | X Gen Us Co. | METHODS AND COMPOSITIONS FOR USE IN PREPARING POLYNUCLEOTIDES |
| US10837047B2 (en) | 2017-10-04 | 2020-11-17 | 10X Genomics, Inc. | Compositions, methods, and systems for bead formation using improved polymers |
| WO2019076768A1 (en) * | 2017-10-16 | 2019-04-25 | Tervisetehnoloogiate Arenduskeskus As | METHOD AND KIT FOR PREPARING DNA BANK |
| WO2019084043A1 (en) | 2017-10-26 | 2019-05-02 | 10X Genomics, Inc. | METHODS AND SYSTEMS FOR NUCLEIC ACID PREPARATION AND CHROMATIN ANALYSIS |
| EP4241882A3 (en) | 2017-10-27 | 2023-12-06 | 10X Genomics, Inc. | Methods for sample preparation and analysis |
| WO2019089836A1 (en) | 2017-10-31 | 2019-05-09 | Encodia, Inc. | Kits for analysis using nucleic acid encoding and/or label |
| WO2019089959A1 (en) | 2017-11-02 | 2019-05-09 | Bio-Rad Laboratories, Inc. | Transposase-based genomic analysis |
| EP3625361A1 (en) | 2017-11-15 | 2020-03-25 | 10X Genomics, Inc. | Functionalized gel beads |
| US10829815B2 (en) | 2017-11-17 | 2020-11-10 | 10X Genomics, Inc. | Methods and systems for associating physical and genetic properties of biological particles |
| WO2019104034A1 (en) | 2017-11-21 | 2019-05-31 | Arima Genomics, Inc. | Preserving spatial-proximal contiguity and molecular contiguity in nucleic acid templates |
| WO2019108851A1 (en) | 2017-11-30 | 2019-06-06 | 10X Genomics, Inc. | Systems and methods for nucleic acid preparation and analysis |
| CN111712579B (zh) | 2017-12-22 | 2024-10-15 | 10X基因组学有限公司 | 用于处理来自一个或多个细胞的核酸分子的系统和方法 |
| WO2019157529A1 (en) | 2018-02-12 | 2019-08-15 | 10X Genomics, Inc. | Methods characterizing multiple analytes from individual cells or cell populations |
| US11639928B2 (en) | 2018-02-22 | 2023-05-02 | 10X Genomics, Inc. | Methods and systems for characterizing analytes from individual cells or cell populations |
| WO2019169028A1 (en) | 2018-02-28 | 2019-09-06 | 10X Genomics, Inc. | Transcriptome sequencing through random ligation |
| WO2019195166A1 (en) | 2018-04-06 | 2019-10-10 | 10X Genomics, Inc. | Systems and methods for quality control in single cell processing |
| CN112272710A (zh) * | 2018-05-03 | 2021-01-26 | 贝克顿迪金森公司 | 高通量多组学样品分析 |
| US11365409B2 (en) | 2018-05-03 | 2022-06-21 | Becton, Dickinson And Company | Molecular barcoding on opposite transcript ends |
| ES2947437T3 (es) | 2018-05-08 | 2023-08-09 | Mgi Tech Co Ltd | Creación de Códigos de barra compartidos en el ADN, en un solo tubo con perlas, para la secuenciación, haplotipado y ensamblaje preciso y rentable |
| WO2019217758A1 (en) | 2018-05-10 | 2019-11-14 | 10X Genomics, Inc. | Methods and systems for molecular library generation |
| CA3067435C (en) * | 2018-05-17 | 2023-09-12 | Illumina, Inc. | High-throughput single-cell sequencing with reduced amplification bias |
| US11932899B2 (en) | 2018-06-07 | 2024-03-19 | 10X Genomics, Inc. | Methods and systems for characterizing nucleic acid molecules |
| US11703427B2 (en) | 2018-06-25 | 2023-07-18 | 10X Genomics, Inc. | Methods and systems for cell and bead processing |
| US20200032335A1 (en) | 2018-07-27 | 2020-01-30 | 10X Genomics, Inc. | Systems and methods for metabolome analysis |
| CN113166807B (zh) | 2018-08-20 | 2024-06-04 | 生物辐射实验室股份有限公司 | 通过分区中条码珠共定位生成核苷酸序列 |
| US12065688B2 (en) | 2018-08-20 | 2024-08-20 | 10X Genomics, Inc. | Compositions and methods for cellular processing |
| EP3859014A4 (en) * | 2018-09-27 | 2022-04-27 | BGI Shenzhen | METHOD OF CONSTRUCTING SEQUENCING LIBRARY, OBTAINED SEQUENCING LIBRARY AND METHOD OF SEQUENCING |
| EP3861134B1 (en) | 2018-10-01 | 2024-09-04 | Becton, Dickinson and Company | Determining 5' transcript sequences |
| SG11202102722SA (en) * | 2018-10-25 | 2021-04-29 | Illumina Inc | Methods and compositions for identifying ligands on arrays using indexes and barcodes |
| US11932849B2 (en) | 2018-11-08 | 2024-03-19 | Becton, Dickinson And Company | Whole transcriptome analysis of single cells using random priming |
| US11459607B1 (en) | 2018-12-10 | 2022-10-04 | 10X Genomics, Inc. | Systems and methods for processing-nucleic acid molecules from a single cell using sequential co-partitioning and composite barcodes |
| EP3894552A1 (en) | 2018-12-13 | 2021-10-20 | Becton, Dickinson and Company | Selective extension in single cell whole transcriptome analysis |
| US11845983B1 (en) | 2019-01-09 | 2023-12-19 | 10X Genomics, Inc. | Methods and systems for multiplexing of droplet based assays |
| CA3114732A1 (en) | 2019-01-11 | 2020-07-16 | Illumina Cambridge Limited | Complex surface-bound transposome complexes |
| US11661631B2 (en) | 2019-01-23 | 2023-05-30 | Becton, Dickinson And Company | Oligonucleotides associated with antibodies |
| CN113366115B (zh) * | 2019-01-29 | 2024-07-05 | 深圳华大智造科技股份有限公司 | 高覆盖率stlfr |
| CA3121456A1 (en) | 2019-01-29 | 2020-08-06 | Illumina, Inc. | Sequencing kits |
| CN112739809B (zh) | 2019-01-29 | 2024-06-28 | 伊鲁米纳公司 | 流动池 |
| TW202100247A (zh) | 2019-01-29 | 2021-01-01 | 美商伊路米納有限公司 | 流通槽 |
| EP3924505A1 (en) | 2019-02-12 | 2021-12-22 | 10X Genomics, Inc. | Methods for processing nucleic acid molecules |
| US11851683B1 (en) | 2019-02-12 | 2023-12-26 | 10X Genomics, Inc. | Methods and systems for selective analysis of cellular samples |
| US11467153B2 (en) | 2019-02-12 | 2022-10-11 | 10X Genomics, Inc. | Methods for processing nucleic acid molecules |
| CN113454234A (zh) | 2019-02-14 | 2021-09-28 | 贝克顿迪金森公司 | 杂合体靶向和全转录物组扩增 |
| EP3924504A1 (en) | 2019-02-14 | 2021-12-22 | Max-Planck-Gesellschaft zur Förderung der Wissenschaften e.V. | Haplotagging - haplotype phasing and single-tube combinatorial barcoding of nucleic acid molecules using bead-immobilized tn5 transposase |
| US11655499B1 (en) | 2019-02-25 | 2023-05-23 | 10X Genomics, Inc. | Detection of sequence elements in nucleic acid molecules |
| SG11202111242PA (en) | 2019-03-11 | 2021-11-29 | 10X Genomics Inc | Systems and methods for processing optically tagged beads |
| CA3138367A1 (en) | 2019-04-30 | 2020-11-05 | Encodia, Inc. | Methods for preparing analytes and related kits |
| US20220127596A1 (en) | 2019-07-12 | 2022-04-28 | lllumina Cambridge Limited | Nucleic Acid Library Preparation Using Electrophoresis |
| SG11202105836XA (en) * | 2019-07-12 | 2021-07-29 | Illumina Cambridge Ltd | Compositions and methods for preparing nucleic acid sequencing libraries using crispr/cas9 immobilized on a solid support |
| US11377655B2 (en) | 2019-07-16 | 2022-07-05 | Pacific Biosciences Of California, Inc. | Synthetic nucleic acids having non-natural structures |
| WO2021016239A1 (en) | 2019-07-22 | 2021-01-28 | Becton, Dickinson And Company | Single cell chromatin immunoprecipitation sequencing assay |
| CN114667353A (zh) * | 2019-08-19 | 2022-06-24 | 通用测序技术公司 | 用于核酸测序用于追踪核酸片段起源的方法和组合物 |
| JP2022549048A (ja) * | 2019-09-20 | 2022-11-24 | イルミナ インコーポレイテッド | インデックス及びバーコードを使用してアレイ上のリガンドを識別するための方法及び組成物 |
| JP7489455B2 (ja) * | 2019-10-25 | 2024-05-23 | チャンピン ナショナル ラボラトリー | 哺乳類dnaのメチル化の検出及び分析 |
| US11773436B2 (en) | 2019-11-08 | 2023-10-03 | Becton, Dickinson And Company | Using random priming to obtain full-length V(D)J information for immune repertoire sequencing |
| ES2944559T3 (es) * | 2019-12-02 | 2023-06-22 | Illumina Cambridge Ltd | Formación de imágenes de grupos basadas en el tiempo de fragmentos de bibliotecas de ADN genómico con la contigüidad preservada |
| MX2021011847A (es) | 2019-12-19 | 2021-11-17 | Illumina Inc | Genotecas de celulas unicas de alto rendimiento y metodos para elaborarlas y usarlas. |
| WO2021146207A1 (en) | 2020-01-13 | 2021-07-22 | Becton, Dickinson And Company | Methods and compositions for quantitation of proteins and rna |
| WO2021163625A1 (en) * | 2020-02-12 | 2021-08-19 | Universal Sequencing Technology Corporation | Methods for intracellular barcoding and spatial barcoding |
| US11211144B2 (en) | 2020-02-18 | 2021-12-28 | Tempus Labs, Inc. | Methods and systems for refining copy number variation in a liquid biopsy assay |
| US11211147B2 (en) | 2020-02-18 | 2021-12-28 | Tempus Labs, Inc. | Estimation of circulating tumor fraction using off-target reads of targeted-panel sequencing |
| US11475981B2 (en) | 2020-02-18 | 2022-10-18 | Tempus Labs, Inc. | Methods and systems for dynamic variant thresholding in a liquid biopsy assay |
| US11851700B1 (en) | 2020-05-13 | 2023-12-26 | 10X Genomics, Inc. | Methods, kits, and compositions for processing extracellular molecules |
| CN115605614A (zh) | 2020-05-14 | 2023-01-13 | 贝克顿迪金森公司(Us) | 用于免疫组库谱分析的引物 |
| US11932901B2 (en) | 2020-07-13 | 2024-03-19 | Becton, Dickinson And Company | Target enrichment using nucleic acid probes for scRNAseq |
| CA3198842A1 (en) * | 2020-10-21 | 2022-04-28 | Illumina, Inc. | Sequencing templates comprising multiple inserts and compositions and methods for improving sequencing throughput |
| US12084715B1 (en) | 2020-11-05 | 2024-09-10 | 10X Genomics, Inc. | Methods and systems for reducing artifactual antisense products |
| CN116635533A (zh) | 2020-11-20 | 2023-08-22 | 贝克顿迪金森公司 | 高表达的蛋白和低表达的蛋白的谱分析 |
| WO2022182682A1 (en) | 2021-02-23 | 2022-09-01 | 10X Genomics, Inc. | Probe-based analysis of nucleic acids and proteins |
| CN112950101B (zh) * | 2021-05-13 | 2021-08-17 | 北京富通东方科技有限公司 | 一种基于产量预测的汽车工厂混流线排产方法 |
| EP4430206A1 (en) | 2021-11-10 | 2024-09-18 | Encodia, Inc. | Methods for barcoding macromolecules in individual cells |
| EP4272764A1 (en) | 2022-05-03 | 2023-11-08 | Scipio Bioscience | Method of complexing biological units with particles |
| WO2023230550A2 (en) * | 2022-05-26 | 2023-11-30 | Illumina, Inc. | Preparation of long read nucleic acid libraries |
| WO2023239907A1 (en) * | 2022-06-09 | 2023-12-14 | The Regents Of The University Of California | Single cell co-sequencing of dna methylation and rna |
| WO2024089953A1 (ja) | 2022-10-27 | 2024-05-02 | 住友化学株式会社 | オリゴヌクレオチドの製造方法 |
| WO2024137703A1 (en) * | 2022-12-20 | 2024-06-27 | Illumina, Inc. | Multivalent assemblies for enhanced target hybridization |
Citations (5)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| RU2252964C2 (ru) * | 1999-11-08 | 2005-05-27 | Эйкен Кагаку Кабусики Кайся | Способ и набор для синтеза нуклеиновой кислоты, имеющей нуклеотидную последовательность, где в одной цепи попеременно связаны комплементарные нуклеотидные последовательности |
| WO2012048341A1 (en) * | 2010-10-08 | 2012-04-12 | President And Fellows Of Harvard College | High-throughput single cell barcoding |
| US20120208705A1 (en) * | 2011-02-10 | 2012-08-16 | Steemers Frank J | Linking sequence reads using paired code tags |
| US20130203605A1 (en) * | 2011-02-02 | 2013-08-08 | University Of Washington Through Its Center For Commercialization | Massively parallel contiguity mapping |
| WO2013131962A1 (en) * | 2012-03-06 | 2013-09-12 | Illumina Cambridge Limited | Improved methods of nucleic acid sequencing |
Family Cites Families (115)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CA1323293C (en) | 1987-12-11 | 1993-10-19 | Keith C. Backman | Assay using template-dependent nucleic acid probe reorganization |
| GB8810400D0 (en) | 1988-05-03 | 1988-06-08 | Southern E | Analysing polynucleotide sequences |
| CA1341584C (en) | 1988-04-06 | 2008-11-18 | Bruce Wallace | Method of amplifying and detecting nucleic acid sequences |
| WO1989009835A1 (en) | 1988-04-08 | 1989-10-19 | The Salk Institute For Biological Studies | Ligase-based amplification method |
| DE68927373T2 (de) | 1988-06-24 | 1997-03-20 | Amgen Inc., Thousand Oaks, Calif. | Verfahren und mittel zum nachweis von nukleinsäuresequenzen |
| US5130238A (en) | 1988-06-24 | 1992-07-14 | Cangene Corporation | Enhanced nucleic acid amplification process |
| WO1990001069A1 (en) | 1988-07-20 | 1990-02-08 | Segev Diagnostics, Inc. | Process for amplifying and detecting nucleic acid sequences |
| US5185243A (en) | 1988-08-25 | 1993-02-09 | Syntex (U.S.A.) Inc. | Method for detection of specific nucleic acid sequences |
| WO1991006678A1 (en) | 1989-10-26 | 1991-05-16 | Sri International | Dna sequencing |
| US5573907A (en) | 1990-01-26 | 1996-11-12 | Abbott Laboratories | Detecting and amplifying target nucleic acids using exonucleolytic activity |
| ATE137269T1 (de) | 1990-01-26 | 1996-05-15 | Abbott Lab | Verbessertes verfahren zur amplifikation von nuklein säurezielsequenz, einsetzbar für die polymerase und ligasekettenreaktion |
| US5223414A (en) | 1990-05-07 | 1993-06-29 | Sri International | Process for nucleic acid hybridization and amplification |
| US5455166A (en) | 1991-01-31 | 1995-10-03 | Becton, Dickinson And Company | Strand displacement amplification |
| DE69531542T2 (de) | 1994-02-07 | 2004-06-24 | Beckman Coulter, Inc., Fullerton | Ligase/polymerase-vermittelte analyse genetischer elemente von einzelnukleotid-polymorphismen und ihre verwendung in der genetischen analyse |
| US5677170A (en) | 1994-03-02 | 1997-10-14 | The Johns Hopkins University | In vitro transposition of artificial transposons |
| KR100230718B1 (ko) | 1994-03-16 | 1999-11-15 | 다니엘 엘. 캐시앙, 헨리 엘. 노르호프 | 등온 가닥 변위 핵산 증폭법 |
| US5552278A (en) | 1994-04-04 | 1996-09-03 | Spectragen, Inc. | DNA sequencing by stepwise ligation and cleavage |
| US5641658A (en) | 1994-08-03 | 1997-06-24 | Mosaic Technologies, Inc. | Method for performing amplification of nucleic acid with two primers bound to a single solid support |
| ES2126332T3 (es) | 1994-12-09 | 1999-03-16 | Imp College Innovations Ltd | Metodo para la identificacion de genes implicados en la adaptacion de un microorganismo a su entorno. |
| US5750341A (en) | 1995-04-17 | 1998-05-12 | Lynx Therapeutics, Inc. | DNA sequencing by parallel oligonucleotide extensions |
| US5965443A (en) | 1996-09-09 | 1999-10-12 | Wisconsin Alumni Research Foundation | System for in vitro transposition |
| US5925545A (en) | 1996-09-09 | 1999-07-20 | Wisconsin Alumni Research Foundation | System for in vitro transposition |
| GB9620209D0 (en) | 1996-09-27 | 1996-11-13 | Cemu Bioteknik Ab | Method of sequencing DNA |
| US5858671A (en) | 1996-11-01 | 1999-01-12 | The University Of Iowa Research Foundation | Iterative and regenerative DNA sequencing method |
| GB9626815D0 (en) | 1996-12-23 | 1997-02-12 | Cemu Bioteknik Ab | Method of sequencing DNA |
| WO1998044152A1 (en) | 1997-04-01 | 1998-10-08 | Glaxo Group Limited | Method of nucleic acid sequencing |
| EP3034626A1 (en) | 1997-04-01 | 2016-06-22 | Illumina Cambridge Limited | Method of nucleic acid sequencing |
| FI103809B (fi) | 1997-07-14 | 1999-09-30 | Finnzymes Oy | In vitro -menetelmä templaattien tuottamiseksi DNA-sekventointia varten |
| AR021833A1 (es) | 1998-09-30 | 2002-08-07 | Applied Research Systems | Metodos de amplificacion y secuenciacion de acido nucleico |
| US20010046669A1 (en) | 1999-02-24 | 2001-11-29 | Mccobmie William R. | Genetically filtered shotgun sequencing of complex eukaryotic genomes |
| US6355431B1 (en) | 1999-04-20 | 2002-03-12 | Illumina, Inc. | Detection of nucleic acid amplification reactions using bead arrays |
| US20050244870A1 (en) | 1999-04-20 | 2005-11-03 | Illumina, Inc. | Nucleic acid sequencing using microsphere arrays |
| US6274320B1 (en) | 1999-09-16 | 2001-08-14 | Curagen Corporation | Method of sequencing a nucleic acid |
| US7244559B2 (en) | 1999-09-16 | 2007-07-17 | 454 Life Sciences Corporation | Method of sequencing a nucleic acid |
| US7582420B2 (en) | 2001-07-12 | 2009-09-01 | Illumina, Inc. | Multiplex nucleic acid reactions |
| US7955794B2 (en) | 2000-09-21 | 2011-06-07 | Illumina, Inc. | Multiplex nucleic acid reactions |
| US7611869B2 (en) | 2000-02-07 | 2009-11-03 | Illumina, Inc. | Multiplexed methylation detection methods |
| US7001792B2 (en) | 2000-04-24 | 2006-02-21 | Eagle Research & Development, Llc | Ultra-fast nucleic acid sequencing device and a method for making and using the same |
| WO2001090415A2 (en) | 2000-05-20 | 2001-11-29 | The Regents Of The University Of Michigan | Method of producing a dna library using positional amplification |
| JP2004513619A (ja) | 2000-07-07 | 2004-05-13 | ヴィジゲン バイオテクノロジーズ インコーポレイテッド | リアルタイム配列決定 |
| US6846658B1 (en) | 2000-10-12 | 2005-01-25 | New England Biolabs, Inc. | Method for cloning and producing the Msel restriction endonuclease |
| AU2002227156A1 (en) | 2000-12-01 | 2002-06-11 | Visigen Biotechnologies, Inc. | Enzymatic nucleic acid synthesis: compositions and methods for altering monomer incorporation fidelity |
| AR031640A1 (es) | 2000-12-08 | 2003-09-24 | Applied Research Systems | Amplificacion isotermica de acidos nucleicos en un soporte solido |
| US20040110191A1 (en) | 2001-01-31 | 2004-06-10 | Winkler Matthew M. | Comparative analysis of nucleic acids using population tagging |
| US7138267B1 (en) | 2001-04-04 | 2006-11-21 | Epicentre Technologies Corporation | Methods and compositions for amplifying DNA clone copy number |
| US6777187B2 (en) | 2001-05-02 | 2004-08-17 | Rubicon Genomics, Inc. | Genome walking by selective amplification of nick-translate DNA library and amplification from complex mixtures of templates |
| GB0115194D0 (en) | 2001-06-21 | 2001-08-15 | Leuven K U Res & Dev | Novel technology for genetic mapping |
| US7057026B2 (en) | 2001-12-04 | 2006-06-06 | Solexa Limited | Labelled nucleotides |
| US7399590B2 (en) | 2002-02-21 | 2008-07-15 | Asm Scientific, Inc. | Recombinase polymerase amplification |
| US20040002090A1 (en) | 2002-03-05 | 2004-01-01 | Pascal Mayer | Methods for detecting genome-wide sequence variations associated with a phenotype |
| DK3363809T3 (da) | 2002-08-23 | 2020-05-04 | Illumina Cambridge Ltd | Modificerede nukleotider til polynukleotidsekvensering |
| US7595883B1 (en) | 2002-09-16 | 2009-09-29 | The Board Of Trustees Of The Leland Stanford Junior University | Biological analysis arrangement and approach therefor |
| AU2003277984A1 (en) | 2002-11-05 | 2004-06-07 | The University Of Queensland | Nucleotide sequence analysis by quantification of mutagenesis |
| US7575865B2 (en) | 2003-01-29 | 2009-08-18 | 454 Life Sciences Corporation | Methods of amplifying and sequencing nucleic acids |
| DK1594971T3 (da) | 2003-02-10 | 2011-05-23 | Max Delbrueck Centrum | Transposonbaseret målretningssystem |
| AU2004253882B2 (en) | 2003-06-20 | 2010-06-10 | Illumina, Inc. | Methods and compositions for whole genome amplification and genotyping |
| US20060024681A1 (en) | 2003-10-31 | 2006-02-02 | Agencourt Bioscience Corporation | Methods for producing a paired tag from a nucleic acid sequence and methods of use thereof |
| EP3175914A1 (en) | 2004-01-07 | 2017-06-07 | Illumina Cambridge Limited | Improvements in or relating to molecular arrays |
| US7595160B2 (en) | 2004-01-13 | 2009-09-29 | U.S. Genomics, Inc. | Analyte detection using barcoded polymers |
| WO2005100585A2 (en) | 2004-03-30 | 2005-10-27 | Epicentre | Methods for obtaining directionally truncated polypeptides |
| WO2006047183A2 (en) | 2004-10-21 | 2006-05-04 | New England Biolabs, Inc. | Recombinant dna nicking endonuclease and uses thereof |
| US7319142B1 (en) | 2004-08-31 | 2008-01-15 | Monsanto Technology Llc | Nucleotide and amino acid sequences from Xenorhabdus and uses thereof |
| AU2005296200B2 (en) | 2004-09-17 | 2011-07-14 | Pacific Biosciences Of California, Inc. | Apparatus and method for analysis of molecules |
| US7449297B2 (en) | 2005-04-14 | 2008-11-11 | Euclid Diagnostics Llc | Methods of copying the methylation pattern of DNA during isothermal amplification and microarrays |
| CA2615323A1 (en) | 2005-06-06 | 2007-12-21 | 454 Life Sciences Corporation | Paired end sequencing |
| EP2463386B1 (en) | 2005-06-15 | 2017-04-12 | Complete Genomics Inc. | Nucleic acid analysis by random mixtures of non-overlapping fragments |
| US7405281B2 (en) | 2005-09-29 | 2008-07-29 | Pacific Biosciences Of California, Inc. | Fluorescent nucleotide analogs and uses therefor |
| GB0522310D0 (en) | 2005-11-01 | 2005-12-07 | Solexa Ltd | Methods of preparing libraries of template polynucleotides |
| US20070128610A1 (en) | 2005-12-02 | 2007-06-07 | Buzby Philip R | Sample preparation method and apparatus for nucleic acid sequencing |
| WO2007087312A2 (en) | 2006-01-23 | 2007-08-02 | Population Genetics Technologies Ltd. | Molecular counting |
| WO2007107710A1 (en) | 2006-03-17 | 2007-09-27 | Solexa Limited | Isothermal methods for creating clonal single molecule arrays |
| EP3722409A1 (en) | 2006-03-31 | 2020-10-14 | Illumina, Inc. | Systems and devices for sequence by synthesis analysis |
| WO2008005459A2 (en) | 2006-06-30 | 2008-01-10 | Nugen Technologies, Inc. | Methods for fragmentation and labeling of nucleic acids |
| US7754429B2 (en) | 2006-10-06 | 2010-07-13 | Illumina Cambridge Limited | Method for pair-wise sequencing a plurity of target polynucleotides |
| CA2666517A1 (en) | 2006-10-23 | 2008-05-02 | Pacific Biosciences Of California, Inc. | Polymerase enzymes and reagents for enhanced nucleic acid sequencing |
| US20090088331A1 (en) | 2006-11-07 | 2009-04-02 | Government Of The United States Of America, As Represented By The Secretary, Dept. Of Health And | Influenza virus nucleic acid microarray and method of use |
| US20080242560A1 (en) | 2006-11-21 | 2008-10-02 | Gunderson Kevin L | Methods for generating amplified nucleic acid arrays |
| US8349167B2 (en) | 2006-12-14 | 2013-01-08 | Life Technologies Corporation | Methods and apparatus for detecting molecular interactions using FET arrays |
| US8262900B2 (en) | 2006-12-14 | 2012-09-11 | Life Technologies Corporation | Methods and apparatus for measuring analytes using large scale FET arrays |
| EP4134667A1 (en) | 2006-12-14 | 2023-02-15 | Life Technologies Corporation | Apparatus for measuring analytes using fet arrays |
| AU2008207110B2 (en) * | 2007-01-19 | 2013-10-17 | Epigenomics Ag | Methods and nucleic acids for analyses of cell proliferative disorders |
| BRPI0813718A2 (pt) | 2007-07-13 | 2014-12-30 | Univ Leland Stanford Junior | Método e aparelho que utilizam um campo elétrico para ensaios biológicos aperfeiçoados |
| EP2188389A4 (en) | 2007-08-15 | 2011-12-07 | Univ Hong Kong | METHOD AND COMPOSITIONS FOR BISULFIT DNA SEQUENCING WITH HIGH PERFORMANCE AND BENEFITS |
| US8415099B2 (en) | 2007-11-05 | 2013-04-09 | Complete Genomics, Inc. | Efficient base determination in sequencing reactions |
| WO2009092035A2 (en) | 2008-01-17 | 2009-07-23 | Sequenom, Inc. | Methods and compositions for the analysis of biological molecules |
| CA2722541C (en) | 2008-04-30 | 2017-01-10 | Integrated Dna Technologies, Inc. | Rnase h-based assays utilizing modified rna monomers |
| KR20110025993A (ko) | 2008-06-30 | 2011-03-14 | 바이오나노매트릭스, 인크. | 단일-분자 전체 게놈 분석용 장치 및 방법 |
| US8383345B2 (en) | 2008-09-12 | 2013-02-26 | University Of Washington | Sequence tag directed subassembly of short sequencing reads into long sequencing reads |
| US20100137143A1 (en) | 2008-10-22 | 2010-06-03 | Ion Torrent Systems Incorporated | Methods and apparatus for measuring analytes |
| US9080211B2 (en) | 2008-10-24 | 2015-07-14 | Epicentre Technologies Corporation | Transposon end compositions and methods for modifying nucleic acids |
| CA2750054C (en) | 2008-10-24 | 2018-05-29 | Epicentre Technologies Corporation | Transposon end compositions and methods for modifying nucleic acids |
| WO2010099301A2 (en) | 2009-02-25 | 2010-09-02 | The Johns Hopkins University | Piggybac transposon variants and methods of use |
| US8709717B2 (en) | 2009-04-03 | 2014-04-29 | Illumina, Inc. | Generation of uniform fragments of nucleic acids using patterned substrates |
| HUE027972T2 (en) | 2010-02-25 | 2016-11-28 | Advanced Liquid Logic Inc | A method for generating nucleic acid libraries |
| CN103080338A (zh) * | 2010-08-27 | 2013-05-01 | 弗·哈夫曼-拉罗切有限公司 | 核酸捕获和测序方法 |
| US9029103B2 (en) | 2010-08-27 | 2015-05-12 | Illumina Cambridge Limited | Methods for sequencing polynucleotides |
| US9096899B2 (en) | 2010-10-27 | 2015-08-04 | Illumina, Inc. | Microdevices and biosensor cartridges for biological or chemical analysis and systems and methods for the same |
| US9074251B2 (en) | 2011-02-10 | 2015-07-07 | Illumina, Inc. | Linking sequence reads using paired code tags |
| EP2635679B1 (en) | 2010-11-05 | 2017-04-19 | Illumina, Inc. | Linking sequence reads using paired code tags |
| US8951781B2 (en) | 2011-01-10 | 2015-02-10 | Illumina, Inc. | Systems, methods, and apparatuses to image a sample for biological or chemical analysis |
| AU2012211081B2 (en) * | 2011-01-28 | 2016-02-04 | Illumina, Inc. | Oligonucleotide replacement for di-tagged and directional libraries |
| US9365897B2 (en) * | 2011-02-08 | 2016-06-14 | Illumina, Inc. | Selective enrichment of nucleic acids |
| AU2012249759A1 (en) | 2011-04-25 | 2013-11-07 | Bio-Rad Laboratories, Inc. | Methods and compositions for nucleic acid analysis |
| US20130017978A1 (en) | 2011-07-11 | 2013-01-17 | Finnzymes Oy | Methods and transposon nucleic acids for generating a dna library |
| AU2013232131B2 (en) | 2012-03-13 | 2018-09-20 | Integrated Dna Technologies, Inc. | Methods and compositions for size-controlled homopolymer tailing of substrate polynucleotides by a nucleic acid polymerase |
| CN104736722B (zh) | 2012-05-21 | 2018-08-07 | 斯克利普斯研究所 | 样品制备方法 |
| US9012022B2 (en) | 2012-06-08 | 2015-04-21 | Illumina, Inc. | Polymer coatings |
| US8895249B2 (en) | 2012-06-15 | 2014-11-25 | Illumina, Inc. | Kinetic exclusion amplification of nucleic acid libraries |
| US9644199B2 (en) | 2012-10-01 | 2017-05-09 | Agilent Technologies, Inc. | Immobilized transposase complexes for DNA fragmentation and tagging |
| US9683230B2 (en) | 2013-01-09 | 2017-06-20 | Illumina Cambridge Limited | Sample preparation on a solid support |
| US9644204B2 (en) | 2013-02-08 | 2017-05-09 | 10X Genomics, Inc. | Partitioning and processing of analytes and other species |
| EP2966179B1 (en) * | 2013-03-07 | 2019-05-08 | Sekisui Medical Co., Ltd. | Method for detecting methylated dna |
| WO2014142850A1 (en) | 2013-03-13 | 2014-09-18 | Illumina, Inc. | Methods and compositions for nucleic acid sequencing |
| GB2546833B (en) | 2013-08-28 | 2018-04-18 | Cellular Res Inc | Microwell for single cell analysis comprising single cell and single bead oligonucleotide capture labels |
| CN114717291A (zh) | 2013-12-30 | 2022-07-08 | 阿特雷卡公司 | 使用核酸条形码分析与单细胞缔合的核酸 |
-
2015
- 2015-10-16 RU RU2019138705A patent/RU2736728C2/ru active
- 2015-10-16 WO PCT/US2015/056040 patent/WO2016061517A2/en active Application Filing
- 2015-10-16 BR BR122021026779-0A patent/BR122021026779B1/pt active IP Right Grant
- 2015-10-16 SG SG11201703139VA patent/SG11201703139VA/en unknown
- 2015-10-16 US US15/519,482 patent/US11873480B2/en active Active
- 2015-10-16 KR KR1020227041250A patent/KR102643955B1/ko active IP Right Grant
- 2015-10-16 RU RU2017116989A patent/RU2709655C2/ru active
- 2015-10-16 AU AU2015331739A patent/AU2015331739B2/en active Active
- 2015-10-16 CA CA2964799A patent/CA2964799A1/en active Pending
- 2015-10-16 KR KR1020177013242A patent/KR102472027B1/ko active IP Right Grant
- 2015-10-16 SG SG10201903408VA patent/SG10201903408VA/en unknown
- 2015-10-16 BR BR122021026781-2A patent/BR122021026781B1/pt active IP Right Grant
- 2015-10-16 IL IL299976A patent/IL299976B1/en unknown
- 2015-10-16 JP JP2017520884A patent/JP6808617B2/ja active Active
-
2017
- 2017-04-13 IL IL251737A patent/IL251737B/en unknown
-
2018
- 2018-10-29 US US16/173,202 patent/US20190048332A1/en not_active Abandoned
-
2020
- 2020-12-09 JP JP2020204004A patent/JP7127104B2/ja active Active
-
2021
- 2021-11-04 IL IL287853A patent/IL287853B2/en unknown
-
2022
- 2022-02-22 AU AU2022201205A patent/AU2022201205A1/en active Pending
- 2022-04-12 US US17/719,276 patent/US20220282242A1/en active Pending
- 2022-08-17 JP JP2022129859A patent/JP7532455B2/ja active Active
Patent Citations (5)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| RU2252964C2 (ru) * | 1999-11-08 | 2005-05-27 | Эйкен Кагаку Кабусики Кайся | Способ и набор для синтеза нуклеиновой кислоты, имеющей нуклеотидную последовательность, где в одной цепи попеременно связаны комплементарные нуклеотидные последовательности |
| WO2012048341A1 (en) * | 2010-10-08 | 2012-04-12 | President And Fellows Of Harvard College | High-throughput single cell barcoding |
| US20130203605A1 (en) * | 2011-02-02 | 2013-08-08 | University Of Washington Through Its Center For Commercialization | Massively parallel contiguity mapping |
| US20120208705A1 (en) * | 2011-02-10 | 2012-08-16 | Steemers Frank J | Linking sequence reads using paired code tags |
| WO2013131962A1 (en) * | 2012-03-06 | 2013-09-12 | Illumina Cambridge Limited | Improved methods of nucleic acid sequencing |
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| RU2736728C2 (ru) | Транспозиция с сохранением сцепления генов | |
| CN107969137B (zh) | 接近性保留性转座 | |
| CN105189308B (zh) | 长dna片段的多重标记 | |
| CN107109485B (zh) | 用于多重捕获反应的通用阻断寡聚物系统和改进的杂交捕获的方法 | |
| CN111201329A (zh) | 具有减少的扩增偏倚的高通量单细胞测序 | |
| JP2020501554A (ja) | 短いdna断片を連結することによる一分子シーケンスのスループットを増加する方法 | |
| US20160348152A1 (en) | Compositions and Methods for Preparing Sequencing Libraries | |
| US20230383336A1 (en) | Method for nucleic acid detection by oligo hybridization and pcr-based amplification | |
| JP6089012B2 (ja) | Dnaメチル化分析方法 | |
| BR112017007912B1 (pt) | Transposon de preservação de contiguidade |