RU2580045C2 - Антитела человека против ангиопоэтин-подобного белка 4 человека - Google Patents
Антитела человека против ангиопоэтин-подобного белка 4 человека Download PDFInfo
- Publication number
- RU2580045C2 RU2580045C2 RU2012131547/10A RU2012131547A RU2580045C2 RU 2580045 C2 RU2580045 C2 RU 2580045C2 RU 2012131547/10 A RU2012131547/10 A RU 2012131547/10A RU 2012131547 A RU2012131547 A RU 2012131547A RU 2580045 C2 RU2580045 C2 RU 2580045C2
- Authority
- RU
- Russia
- Prior art keywords
- ser
- gly
- thr
- tyr
- antibody
- Prior art date
Links
- 101000693076 Homo sapiens Angiopoietin-related protein 4 Proteins 0.000 title claims abstract description 118
- 102100025674 Angiopoietin-related protein 4 Human genes 0.000 title 1
- 239000012634 fragment Substances 0.000 claims abstract description 113
- 230000027455 binding Effects 0.000 claims abstract description 107
- 239000000427 antigen Substances 0.000 claims abstract description 92
- 102000036639 antigens Human genes 0.000 claims abstract description 92
- 108091007433 antigens Proteins 0.000 claims abstract description 92
- 102000045205 Angiopoietin-Like Protein 4 Human genes 0.000 claims abstract description 87
- 108700042530 Angiopoietin-Like Protein 4 Proteins 0.000 claims abstract description 87
- 101150054149 ANGPTL4 gene Proteins 0.000 claims abstract description 81
- 241000282414 Homo sapiens Species 0.000 claims abstract description 47
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 claims abstract description 47
- 230000000694 effects Effects 0.000 claims abstract description 41
- 238000000034 method Methods 0.000 claims abstract description 39
- 201000010099 disease Diseases 0.000 claims abstract description 25
- 150000007523 nucleic acids Chemical class 0.000 claims abstract description 23
- 208000035475 disorder Diseases 0.000 claims abstract description 22
- 239000003814 drug Substances 0.000 claims abstract description 21
- 239000008194 pharmaceutical composition Substances 0.000 claims abstract description 21
- 108020004707 nucleic acids Proteins 0.000 claims abstract description 20
- 102000039446 nucleic acids Human genes 0.000 claims abstract description 20
- 208000017170 Lipid metabolism disease Diseases 0.000 claims abstract description 11
- 208000032928 Dyslipidaemia Diseases 0.000 claims abstract description 10
- 230000002401 inhibitory effect Effects 0.000 claims abstract description 10
- 208000024172 Cardiovascular disease Diseases 0.000 claims abstract description 9
- 208000006575 hypertriglyceridemia Diseases 0.000 claims abstract description 8
- 230000002829 reductive effect Effects 0.000 claims abstract description 8
- 206010012601 diabetes mellitus Diseases 0.000 claims abstract description 7
- 208000008338 non-alcoholic fatty liver disease Diseases 0.000 claims abstract description 7
- 206010053219 non-alcoholic steatohepatitis Diseases 0.000 claims abstract description 7
- 208000035150 Hypercholesterolemia Diseases 0.000 claims abstract description 6
- 208000008589 Obesity Diseases 0.000 claims abstract description 6
- 235000020824 obesity Nutrition 0.000 claims abstract description 6
- 206010033645 Pancreatitis Diseases 0.000 claims abstract description 5
- 206010033647 Pancreatitis acute Diseases 0.000 claims abstract description 5
- 201000003229 acute pancreatitis Diseases 0.000 claims abstract description 5
- 239000013604 expression vector Substances 0.000 claims abstract description 5
- 238000004519 manufacturing process Methods 0.000 claims abstract description 3
- 125000003275 alpha amino acid group Chemical group 0.000 claims description 71
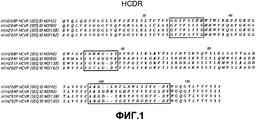
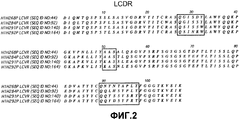
- 108010047041 Complementarity Determining Regions Proteins 0.000 claims description 43
- HVYWMOMLDIMFJA-DPAQBDIFSA-N cholesterol Chemical compound C1C=C2C[C@@H](O)CC[C@]2(C)[C@@H]2[C@@H]1[C@@H]1CC[C@H]([C@H](C)CCCC(C)C)[C@@]1(C)CC2 HVYWMOMLDIMFJA-DPAQBDIFSA-N 0.000 claims description 20
- 239000003795 chemical substances by application Substances 0.000 claims description 14
- 229940124597 therapeutic agent Drugs 0.000 claims description 13
- 241000282560 Macaca mulatta Species 0.000 claims description 11
- 235000012000 cholesterol Nutrition 0.000 claims description 10
- 239000003112 inhibitor Substances 0.000 claims description 9
- 230000001404 mediated effect Effects 0.000 claims description 8
- 230000009260 cross reactivity Effects 0.000 claims description 7
- 241000282567 Macaca fascicularis Species 0.000 claims description 6
- 229940121710 HMGCoA reductase inhibitor Drugs 0.000 claims description 5
- PVNIIMVLHYAWGP-UHFFFAOYSA-N Niacin Chemical compound OC(=O)C1=CC=CN=C1 PVNIIMVLHYAWGP-UHFFFAOYSA-N 0.000 claims description 5
- 230000000923 atherogenic effect Effects 0.000 claims description 5
- 229960003512 nicotinic acid Drugs 0.000 claims description 5
- 235000001968 nicotinic acid Nutrition 0.000 claims description 5
- 239000011664 nicotinic acid Substances 0.000 claims description 5
- 102000004895 Lipoproteins Human genes 0.000 claims description 4
- 108090001030 Lipoproteins Proteins 0.000 claims description 4
- 230000015556 catabolic process Effects 0.000 claims description 4
- 239000012190 activator Substances 0.000 claims description 3
- 229940125753 fibrate Drugs 0.000 claims description 3
- 208000010125 myocardial infarction Diseases 0.000 claims description 3
- HSINOMROUCMIEA-FGVHQWLLSA-N (2s,4r)-4-[(3r,5s,6r,7r,8s,9s,10s,13r,14s,17r)-6-ethyl-3,7-dihydroxy-10,13-dimethyl-2,3,4,5,6,7,8,9,11,12,14,15,16,17-tetradecahydro-1h-cyclopenta[a]phenanthren-17-yl]-2-methylpentanoic acid Chemical compound C([C@@]12C)C[C@@H](O)C[C@H]1[C@@H](CC)[C@@H](O)[C@@H]1[C@@H]2CC[C@]2(C)[C@@H]([C@H](C)C[C@H](C)C(O)=O)CC[C@H]21 HSINOMROUCMIEA-FGVHQWLLSA-N 0.000 claims description 2
- 239000003613 bile acid Substances 0.000 claims description 2
- 239000003937 drug carrier Substances 0.000 claims description 2
- 239000002471 hydroxymethylglutaryl coenzyme A reductase inhibitor Substances 0.000 claims description 2
- 230000009103 reabsorption Effects 0.000 claims description 2
- 230000001476 alcoholic effect Effects 0.000 claims 2
- 102000040945 Transcription factor Human genes 0.000 claims 1
- 108091023040 Transcription factor Proteins 0.000 claims 1
- 230000002265 prevention Effects 0.000 claims 1
- 102000055659 human ANGPTL4 Human genes 0.000 abstract description 12
- 239000000126 substance Substances 0.000 abstract description 11
- 208000031226 Hyperlipidaemia Diseases 0.000 abstract description 7
- 230000037356 lipid metabolism Effects 0.000 abstract description 5
- 208000020346 hyperlipoproteinemia Diseases 0.000 abstract description 4
- 201000001320 Atherosclerosis Diseases 0.000 abstract description 2
- 239000002253 acid Substances 0.000 abstract description 2
- 230000003143 atherosclerotic effect Effects 0.000 abstract 1
- 208000019622 heart disease Diseases 0.000 abstract 1
- 108090000623 proteins and genes Proteins 0.000 description 246
- 235000018102 proteins Nutrition 0.000 description 240
- 102000004169 proteins and genes Human genes 0.000 description 240
- 108020004414 DNA Proteins 0.000 description 221
- FQPDRTDDEZXCEC-SVSWQMSJSA-N Thr-Ile-Ser Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CO)C(O)=O FQPDRTDDEZXCEC-SVSWQMSJSA-N 0.000 description 85
- 150000003626 triacylglycerols Chemical class 0.000 description 80
- QYSFWUIXDFJUDW-DCAQKATOSA-N Ser-Leu-Arg Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O QYSFWUIXDFJUDW-DCAQKATOSA-N 0.000 description 71
- 108010003137 tyrosyltyrosine Proteins 0.000 description 65
- WNZOCXUOGVYYBJ-CDMKHQONSA-N Gly-Phe-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CC1=CC=CC=C1)NC(=O)CN)O WNZOCXUOGVYYBJ-CDMKHQONSA-N 0.000 description 64
- 241000699670 Mus sp. Species 0.000 description 62
- XKUKSGPZAADMRA-UHFFFAOYSA-N glycyl-glycyl-glycine Natural products NCC(=O)NCC(=O)NCC(O)=O XKUKSGPZAADMRA-UHFFFAOYSA-N 0.000 description 61
- UIGMAMGZOJVTDN-WHFBIAKZSA-N Ser-Gly-Ser Chemical compound OC[C@H](N)C(=O)NCC(=O)N[C@@H](CO)C(O)=O UIGMAMGZOJVTDN-WHFBIAKZSA-N 0.000 description 57
- PDUHNKAFQXQNLH-ZETCQYMHSA-N Gly-Lys-Gly Chemical compound NCCCC[C@H](NC(=O)CN)C(=O)NCC(O)=O PDUHNKAFQXQNLH-ZETCQYMHSA-N 0.000 description 53
- FEHQLKKBVJHSEC-SZMVWBNQSA-N Leu-Glu-Trp Chemical compound C1=CC=C2C(C[C@H](NC(=O)[C@H](CCC(O)=O)NC(=O)[C@@H](N)CC(C)C)C(O)=O)=CNC2=C1 FEHQLKKBVJHSEC-SZMVWBNQSA-N 0.000 description 53
- PDIDTSZKKFEDMB-UWVGGRQHSA-N Lys-Pro-Gly Chemical compound [H]N[C@@H](CCCCN)C(=O)N1CCC[C@H]1C(=O)NCC(O)=O PDIDTSZKKFEDMB-UWVGGRQHSA-N 0.000 description 53
- PMGDADKJMCOXHX-UHFFFAOYSA-N L-Arginyl-L-glutamin-acetat Natural products NC(=N)NCCCC(N)C(=O)NC(CCC(N)=O)C(O)=O PMGDADKJMCOXHX-UHFFFAOYSA-N 0.000 description 52
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 description 52
- 108010008355 arginyl-glutamine Proteins 0.000 description 52
- KIZIOFNVSOSKJI-CIUDSAMLSA-N Leu-Ser-Cys Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CS)C(=O)O)N KIZIOFNVSOSKJI-CIUDSAMLSA-N 0.000 description 51
- YMTLKLXDFCSCNX-BYPYZUCNSA-N Ser-Gly-Gly Chemical compound OC[C@H](N)C(=O)NCC(=O)NCC(O)=O YMTLKLXDFCSCNX-BYPYZUCNSA-N 0.000 description 51
- MBLJBGZWLHTJBH-SZMVWBNQSA-N Trp-Val-Arg Chemical compound C1=CC=C2C(C[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCCN=C(N)N)C(O)=O)=CNC2=C1 MBLJBGZWLHTJBH-SZMVWBNQSA-N 0.000 description 51
- KLYYKKGCPOGDPE-OEAJRASXSA-N Phe-Thr-Leu Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(C)C)C(O)=O KLYYKKGCPOGDPE-OEAJRASXSA-N 0.000 description 50
- SZQCDCKIGWQAQN-FXQIFTODSA-N Cys-Arg-Ala Chemical compound [H]N[C@@H](CS)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](C)C(O)=O SZQCDCKIGWQAQN-FXQIFTODSA-N 0.000 description 49
- 108010067216 glycyl-glycyl-glycine Proteins 0.000 description 49
- AOHKLEBWKMKITA-IHRRRGAJSA-N Arg-Phe-Ser Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CO)C(=O)O)NC(=O)[C@H](CCCN=C(N)N)N AOHKLEBWKMKITA-IHRRRGAJSA-N 0.000 description 48
- GJFYFGOEWLDQGW-GUBZILKMSA-N Ser-Leu-Gln Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)O)NC(=O)[C@H](CO)N GJFYFGOEWLDQGW-GUBZILKMSA-N 0.000 description 48
- 108010086434 alanyl-seryl-glycine Proteins 0.000 description 48
- 108010044292 tryptophyltyrosine Proteins 0.000 description 48
- ARHJJAAWNWOACN-FXQIFTODSA-N Ala-Ser-Val Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(O)=O ARHJJAAWNWOACN-FXQIFTODSA-N 0.000 description 47
- XYBJLTKSGFBLCS-QXEWZRGKSA-N Asp-Arg-Val Chemical compound NC(N)=NCCC[C@@H](C(=O)N[C@@H](C(C)C)C(O)=O)NC(=O)[C@@H](N)CC(O)=O XYBJLTKSGFBLCS-QXEWZRGKSA-N 0.000 description 47
- AZWNCEBQZXELEZ-FXQIFTODSA-N Ser-Pro-Ser Chemical compound OC[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CO)C(O)=O AZWNCEBQZXELEZ-FXQIFTODSA-N 0.000 description 47
- SSYBNWFXCFNRFN-GUBZILKMSA-N Val-Pro-Ser Chemical compound CC(C)[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CO)C(O)=O SSYBNWFXCFNRFN-GUBZILKMSA-N 0.000 description 47
- 108010015792 glycyllysine Proteins 0.000 description 47
- 210000002966 serum Anatomy 0.000 description 47
- FQCILXROGNOZON-YUMQZZPRSA-N Gln-Pro-Gly Chemical compound NC(=O)CC[C@H](N)C(=O)N1CCC[C@H]1C(=O)NCC(O)=O FQCILXROGNOZON-YUMQZZPRSA-N 0.000 description 46
- QOIKZODVIPOPDD-AVGNSLFASA-N Tyr-Cys-Gln Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CS)C(=O)N[C@@H](CCC(N)=O)C(O)=O QOIKZODVIPOPDD-AVGNSLFASA-N 0.000 description 46
- YYSWCHMLFJLLBJ-ZLUOBGJFSA-N Ala-Ala-Ser Chemical compound C[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@@H](CO)C(O)=O YYSWCHMLFJLLBJ-ZLUOBGJFSA-N 0.000 description 45
- CQGSYZCULZMEDE-UHFFFAOYSA-N Leu-Gln-Pro Natural products CC(C)CC(N)C(=O)NC(CCC(N)=O)C(=O)N1CCCC1C(O)=O CQGSYZCULZMEDE-UHFFFAOYSA-N 0.000 description 45
- SPSSJSICDYYTQN-HJGDQZAQSA-N Met-Thr-Gln Chemical compound CSCC[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@H](C(O)=O)CCC(N)=O SPSSJSICDYYTQN-HJGDQZAQSA-N 0.000 description 45
- GXUWHVZYDAHFSV-FLBSBUHZSA-N Thr-Ile-Thr Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H]([C@@H](C)O)C(O)=O GXUWHVZYDAHFSV-FLBSBUHZSA-N 0.000 description 45
- QUILOGWWLXMSAT-IHRRRGAJSA-N Tyr-Gln-Gln Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCC(N)=O)C(O)=O QUILOGWWLXMSAT-IHRRRGAJSA-N 0.000 description 45
- HXWALXSAVBLTPK-NUTKFTJISA-N Leu-Ala-Trp Chemical compound C[C@@H](C(=O)N[C@@H](CC1=CNC2=CC=CC=C21)C(=O)O)NC(=O)[C@H](CC(C)C)N HXWALXSAVBLTPK-NUTKFTJISA-N 0.000 description 44
- IXHKPDJKKCUKHS-GARJFASQSA-N Lys-Ala-Pro Chemical compound C[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CCCCN)N IXHKPDJKKCUKHS-GARJFASQSA-N 0.000 description 44
- CREYEAPXISDKSB-FQPOAREZSA-N Ala-Thr-Tyr Chemical compound [H]N[C@@H](C)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O CREYEAPXISDKSB-FQPOAREZSA-N 0.000 description 43
- YOOAQCZYZHGUAZ-KATARQTJSA-N Thr-Leu-Ser Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(O)=O YOOAQCZYZHGUAZ-KATARQTJSA-N 0.000 description 43
- 108010089804 glycyl-threonine Proteins 0.000 description 43
- 108010037850 glycylvaline Proteins 0.000 description 43
- QNFRBNZGVVKBNJ-PEFMBERDSA-N Asp-Ile-Gln Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)O)NC(=O)[C@H](CC(=O)O)N QNFRBNZGVVKBNJ-PEFMBERDSA-N 0.000 description 42
- AIMGJYMCTAABEN-GVXVVHGQSA-N Leu-Val-Glu Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(O)=O)C(O)=O AIMGJYMCTAABEN-GVXVVHGQSA-N 0.000 description 42
- XMBSYZWANAQXEV-UHFFFAOYSA-N N-alpha-L-glutamyl-L-phenylalanine Natural products OC(=O)CCC(N)C(=O)NC(C(O)=O)CC1=CC=CC=C1 XMBSYZWANAQXEV-UHFFFAOYSA-N 0.000 description 42
- BIBYEFRASCNLAA-CDMKHQONSA-N Thr-Phe-Gly Chemical compound C[C@@H](O)[C@H](N)C(=O)N[C@H](C(=O)NCC(O)=O)CC1=CC=CC=C1 BIBYEFRASCNLAA-CDMKHQONSA-N 0.000 description 41
- 235000001014 amino acid Nutrition 0.000 description 41
- MIIVFRCYJABHTQ-ONGXEEELSA-N Gly-Leu-Val Chemical compound [H]NCC(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](C(C)C)C(O)=O MIIVFRCYJABHTQ-ONGXEEELSA-N 0.000 description 40
- DBUNZBWUWCIELX-JHEQGTHGSA-N Gly-Thr-Glu Chemical compound [H]NCC(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCC(O)=O)C(O)=O DBUNZBWUWCIELX-JHEQGTHGSA-N 0.000 description 40
- JXFLPKSDLDEOQK-JHEQGTHGSA-N Gln-Gly-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)CNC(=O)[C@@H](N)CCC(N)=O JXFLPKSDLDEOQK-JHEQGTHGSA-N 0.000 description 37
- MCIXMYKSPQUMJG-SRVKXCTJSA-N Phe-Ser-Ser Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CO)C(=O)N[C@@H](CO)C(O)=O MCIXMYKSPQUMJG-SRVKXCTJSA-N 0.000 description 36
- 108010089975 arginyl-glycyl-aspartyl-serine Proteins 0.000 description 36
- YQPFCZVKMUVZIN-AUTRQRHGSA-N Glu-Val-Gln Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(N)=O)C(O)=O YQPFCZVKMUVZIN-AUTRQRHGSA-N 0.000 description 35
- BYYNJRSNDARRBX-YFKPBYRVSA-N Gly-Gln-Gly Chemical compound NCC(=O)N[C@@H](CCC(N)=O)C(=O)NCC(O)=O BYYNJRSNDARRBX-YFKPBYRVSA-N 0.000 description 35
- MNYNCKZAEIAONY-XGEHTFHBSA-N Thr-Val-Ser Chemical compound C[C@@H](O)[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CO)C(O)=O MNYNCKZAEIAONY-XGEHTFHBSA-N 0.000 description 35
- UGCIQUYEJIEHKX-GVXVVHGQSA-N Lys-Val-Glu Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(O)=O)C(O)=O UGCIQUYEJIEHKX-GVXVVHGQSA-N 0.000 description 34
- 108010020755 prolyl-glycyl-glycine Proteins 0.000 description 34
- FMDHKPRACUXATF-ACZMJKKPSA-N Ser-Gln-Ser Chemical compound OC[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CO)C(O)=O FMDHKPRACUXATF-ACZMJKKPSA-N 0.000 description 33
- KRRMJKMGWWXWDW-STQMWFEESA-N Gly-Arg-Phe Chemical compound NC(=N)NCCC[C@H](NC(=O)CN)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 KRRMJKMGWWXWDW-STQMWFEESA-N 0.000 description 32
- 108010047495 alanylglycine Proteins 0.000 description 32
- 238000006467 substitution reaction Methods 0.000 description 32
- MNQMTYSEKZHIDF-GCJQMDKQSA-N Asp-Thr-Ala Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C)C(O)=O MNQMTYSEKZHIDF-GCJQMDKQSA-N 0.000 description 31
- 108010010147 glycylglutamine Proteins 0.000 description 31
- 108010073969 valyllysine Proteins 0.000 description 31
- SVFOIXMRMLROHO-SRVKXCTJSA-N Asp-Asp-Phe Chemical compound OC(=O)C[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 SVFOIXMRMLROHO-SRVKXCTJSA-N 0.000 description 30
- HTSSXFASOUSJQG-IHPCNDPISA-N Asp-Tyr-Trp Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(O)=O HTSSXFASOUSJQG-IHPCNDPISA-N 0.000 description 30
- XFQOQUWGVCVYON-DCAQKATOSA-N Asp-Met-His Chemical compound OC(=O)C[C@H](N)C(=O)N[C@@H](CCSC)C(=O)N[C@H](C(O)=O)CC1=CN=CN1 XFQOQUWGVCVYON-DCAQKATOSA-N 0.000 description 28
- PMNHJLASAAWELO-FOHZUACHSA-N Gly-Asp-Thr Chemical compound [H]NCC(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O PMNHJLASAAWELO-FOHZUACHSA-N 0.000 description 28
- NNRFRJQMBSBXGO-CIUDSAMLSA-N (3s)-3-[[2-[[(2s)-2-amino-5-(diaminomethylideneamino)pentanoyl]amino]acetyl]amino]-4-[[(1s)-1-carboxy-2-hydroxyethyl]amino]-4-oxobutanoic acid Chemical compound NC(N)=NCCC[C@H](N)C(=O)NCC(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CO)C(O)=O NNRFRJQMBSBXGO-CIUDSAMLSA-N 0.000 description 27
- SQIARYGNVQWOSB-BZSNNMDCSA-N Asp-Tyr-Phe Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O SQIARYGNVQWOSB-BZSNNMDCSA-N 0.000 description 27
- MPLOSMWGDNJSEV-WHFBIAKZSA-N Ala-Gly-Asp Chemical compound [H]N[C@@H](C)C(=O)NCC(=O)N[C@@H](CC(O)=O)C(O)=O MPLOSMWGDNJSEV-WHFBIAKZSA-N 0.000 description 26
- RKRSYHCNPFGMTA-CIUDSAMLSA-N Arg-Glu-Asn Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(N)=O)C(O)=O RKRSYHCNPFGMTA-CIUDSAMLSA-N 0.000 description 26
- OYTPNWYZORARHL-XHNCKOQMSA-N Gln-Ala-Pro Chemical compound C[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CCC(=O)N)N OYTPNWYZORARHL-XHNCKOQMSA-N 0.000 description 26
- BMKNXTJLHFIAAH-CIUDSAMLSA-N Ser-Ser-Leu Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(O)=O BMKNXTJLHFIAAH-CIUDSAMLSA-N 0.000 description 26
- KHPLUFDSWGDRHD-SLFFLAALSA-N Tyr-Tyr-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CC2=CC=C(C=C2)O)NC(=O)[C@H](CC3=CC=C(C=C3)O)N)C(=O)O KHPLUFDSWGDRHD-SLFFLAALSA-N 0.000 description 26
- WTWGOQRNRFHFQD-JBDRJPRFSA-N Ser-Ala-Ile Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](C)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O WTWGOQRNRFHFQD-JBDRJPRFSA-N 0.000 description 25
- KZSYAEWQMJEGRZ-RHYQMDGZSA-N Thr-Leu-Val Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](C(C)C)C(O)=O KZSYAEWQMJEGRZ-RHYQMDGZSA-N 0.000 description 25
- 108010068265 aspartyltyrosine Proteins 0.000 description 25
- AJBVYEYZVYPFCF-CIUDSAMLSA-N Ala-Lys-Asn Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(N)=O)C(O)=O AJBVYEYZVYPFCF-CIUDSAMLSA-N 0.000 description 24
- JAGGEZACYAAMIL-CQDKDKBSSA-N Tyr-Lys-Ala Chemical compound C[C@@H](C(=O)O)NC(=O)[C@H](CCCCN)NC(=O)[C@H](CC1=CC=C(C=C1)O)N JAGGEZACYAAMIL-CQDKDKBSSA-N 0.000 description 24
- 150000001413 amino acids Chemical class 0.000 description 24
- 238000002347 injection Methods 0.000 description 24
- 239000007924 injection Substances 0.000 description 24
- 108010013563 Lipoprotein Lipase Proteins 0.000 description 23
- 102000043296 Lipoprotein lipases Human genes 0.000 description 23
- 229940024606 amino acid Drugs 0.000 description 23
- 210000004602 germ cell Anatomy 0.000 description 23
- MKJBPDLENBUHQU-CIUDSAMLSA-N Asn-Ser-Leu Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(O)=O MKJBPDLENBUHQU-CIUDSAMLSA-N 0.000 description 22
- MSHXWFKYXJTLEZ-CIUDSAMLSA-N Gln-Met-Asn Chemical compound CSCC[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)O)NC(=O)[C@H](CCC(=O)N)N MSHXWFKYXJTLEZ-CIUDSAMLSA-N 0.000 description 22
- JWOBLHJRDADHLN-KKUMJFAQSA-N Ser-Leu-Tyr Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O JWOBLHJRDADHLN-KKUMJFAQSA-N 0.000 description 22
- ANHVRCNNGJMJNG-BZSNNMDCSA-N Tyr-Tyr-Cys Chemical compound C1=CC(=CC=C1C[C@@H](C(=O)N[C@@H](CC2=CC=C(C=C2)O)C(=O)N[C@@H](CS)C(=O)O)N)O ANHVRCNNGJMJNG-BZSNNMDCSA-N 0.000 description 22
- 108010069020 alanyl-prolyl-glycine Proteins 0.000 description 22
- OTUQSEPIIVBYEM-IHRRRGAJSA-N Arg-Asn-Tyr Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O OTUQSEPIIVBYEM-IHRRRGAJSA-N 0.000 description 21
- RFTVTKBHDXCEEX-WDSKDSINSA-N Glu-Ser-Gly Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CO)C(=O)NCC(O)=O RFTVTKBHDXCEEX-WDSKDSINSA-N 0.000 description 20
- XSLXHSYIVPGEER-KZVJFYERSA-N Thr-Ala-Val Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C)C(=O)N[C@@H](C(C)C)C(O)=O XSLXHSYIVPGEER-KZVJFYERSA-N 0.000 description 20
- 108010009962 valyltyrosine Proteins 0.000 description 20
- OSCLNNWLKKIQJM-WDSKDSINSA-N Gln-Ser-Gly Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CO)C(=O)NCC(O)=O OSCLNNWLKKIQJM-WDSKDSINSA-N 0.000 description 19
- 241000880493 Leptailurus serval Species 0.000 description 19
- 108090000765 processed proteins & peptides Proteins 0.000 description 19
- WPJDPEOQUIXXOY-AVGNSLFASA-N Gln-Tyr-Asn Chemical compound C1=CC(=CC=C1C[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)O)NC(=O)[C@H](CCC(=O)N)N)O WPJDPEOQUIXXOY-AVGNSLFASA-N 0.000 description 18
- ZFBBMCKQSNJZSN-AUTRQRHGSA-N Gln-Val-Gln Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(N)=O)C(O)=O ZFBBMCKQSNJZSN-AUTRQRHGSA-N 0.000 description 18
- ZLCLYFGMKFCDCN-XPUUQOCRSA-N Gly-Ser-Val Chemical compound CC(C)[C@H](NC(=O)[C@H](CO)NC(=O)CN)C(O)=O ZLCLYFGMKFCDCN-XPUUQOCRSA-N 0.000 description 18
- AXZGZMGRBDQTEY-SRVKXCTJSA-N Leu-Gln-Met Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCSC)C(O)=O AXZGZMGRBDQTEY-SRVKXCTJSA-N 0.000 description 18
- FGWUALWGCZJQDJ-URLPEUOOSA-N Phe-Thr-Ile Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O FGWUALWGCZJQDJ-URLPEUOOSA-N 0.000 description 18
- QEDMOZUJTGEIBF-FXQIFTODSA-N Ser-Arg-Asp Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(O)=O)C(O)=O QEDMOZUJTGEIBF-FXQIFTODSA-N 0.000 description 18
- 108010029020 prolylglycine Proteins 0.000 description 18
- MTCFGRXMJLQNBG-REOHCLBHSA-N (2S)-2-Amino-3-hydroxypropansäure Chemical compound OC[C@H](N)C(O)=O MTCFGRXMJLQNBG-REOHCLBHSA-N 0.000 description 17
- PAQUJCSYVIBPLC-AVGNSLFASA-N Glu-Asp-Phe Chemical compound OC(=O)CC[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 PAQUJCSYVIBPLC-AVGNSLFASA-N 0.000 description 17
- 241001465754 Metazoa Species 0.000 description 17
- 108010061238 threonyl-glycine Proteins 0.000 description 17
- FFJQHWKSGAWSTJ-BFHQHQDPSA-N Gly-Thr-Ala Chemical compound [H]NCC(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C)C(O)=O FFJQHWKSGAWSTJ-BFHQHQDPSA-N 0.000 description 16
- GQZMPWBZQALKJO-UWVGGRQHSA-N Lys-Gly-Arg Chemical compound [H]N[C@@H](CCCCN)C(=O)NCC(=O)N[C@@H](CCCNC(N)=N)C(O)=O GQZMPWBZQALKJO-UWVGGRQHSA-N 0.000 description 16
- AIRZWUMAHCDDHR-KKUMJFAQSA-N Lys-Leu-Leu Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC(C)C)C(O)=O AIRZWUMAHCDDHR-KKUMJFAQSA-N 0.000 description 16
- DRRXXZBXDMLGFC-IHRRRGAJSA-N Lys-Val-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](C(C)C)NC(=O)[C@@H](N)CCCCN DRRXXZBXDMLGFC-IHRRRGAJSA-N 0.000 description 16
- OWFGFHQMSBTKLX-UFYCRDLUSA-N Val-Tyr-Tyr Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)N[C@@H](CC2=CC=C(C=C2)O)C(=O)O)N OWFGFHQMSBTKLX-UFYCRDLUSA-N 0.000 description 16
- 150000002632 lipids Chemical class 0.000 description 16
- 239000000203 mixture Substances 0.000 description 16
- VVKVHAOOUGNDPJ-SRVKXCTJSA-N Ser-Tyr-Ser Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CO)C(O)=O VVKVHAOOUGNDPJ-SRVKXCTJSA-N 0.000 description 15
- NWEGIYMHTZXVBP-JSGCOSHPSA-N Tyr-Val-Gly Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](C(C)C)C(=O)NCC(O)=O NWEGIYMHTZXVBP-JSGCOSHPSA-N 0.000 description 15
- 230000005764 inhibitory process Effects 0.000 description 15
- DHMQDGOQFOQNFH-UHFFFAOYSA-N Glycine Chemical compound NCC(O)=O DHMQDGOQFOQNFH-UHFFFAOYSA-N 0.000 description 14
- PXKACEXYLPBMAD-JBDRJPRFSA-N Ile-Ser-Ser Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CO)C(=O)O)N PXKACEXYLPBMAD-JBDRJPRFSA-N 0.000 description 14
- 241000699666 Mus <mouse, genus> Species 0.000 description 14
- XWCYBVBLJRWOFR-WDSKDSINSA-N Ser-Gln-Gly Chemical compound OC[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(=O)NCC(O)=O XWCYBVBLJRWOFR-WDSKDSINSA-N 0.000 description 14
- VCXWRWYFJLXITF-AUTRQRHGSA-N Tyr-Ala-Ala Chemical compound OC(=O)[C@H](C)NC(=O)[C@H](C)NC(=O)[C@@H](N)CC1=CC=C(O)C=C1 VCXWRWYFJLXITF-AUTRQRHGSA-N 0.000 description 14
- 108091028043 Nucleic acid sequence Proteins 0.000 description 13
- PZTZYZUTCPZWJH-FXQIFTODSA-N Val-Ser-Ser Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CO)C(=O)O)N PZTZYZUTCPZWJH-FXQIFTODSA-N 0.000 description 13
- 238000004458 analytical method Methods 0.000 description 13
- 108010069205 aspartyl-phenylalanine Proteins 0.000 description 13
- BUDNAJYVCUHLSV-ZLUOBGJFSA-N Ala-Asp-Ser Chemical compound C[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CO)C(O)=O BUDNAJYVCUHLSV-ZLUOBGJFSA-N 0.000 description 12
- SDZRIBWEVVRDQI-CIUDSAMLSA-N Ala-Lys-Asp Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(O)=O)C(O)=O SDZRIBWEVVRDQI-CIUDSAMLSA-N 0.000 description 12
- UGXYFDQFLVCDFC-CIUDSAMLSA-N Asn-Ser-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CC(N)=O UGXYFDQFLVCDFC-CIUDSAMLSA-N 0.000 description 12
- JBDLMLZNDRLDIX-HJGDQZAQSA-N Asn-Thr-Leu Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(C)C)C(O)=O JBDLMLZNDRLDIX-HJGDQZAQSA-N 0.000 description 12
- XXLBHPPXDUWYAG-XQXXSGGOSA-N Gln-Ala-Thr Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O XXLBHPPXDUWYAG-XQXXSGGOSA-N 0.000 description 12
- LOJYQMFIIJVETK-WDSKDSINSA-N Gln-Gln Chemical compound NC(=O)CC[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(O)=O LOJYQMFIIJVETK-WDSKDSINSA-N 0.000 description 12
- JHNJNTMTZHEDLJ-NAKRPEOUSA-N Ile-Ser-Arg Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H](CO)C(=O)N[C@@H](CCCN=C(N)N)C(O)=O JHNJNTMTZHEDLJ-NAKRPEOUSA-N 0.000 description 12
- IAOZOFPONWDXNT-IXOXFDKPSA-N Phe-Ser-Thr Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CO)C(=O)N[C@@H]([C@@H](C)O)C(O)=O IAOZOFPONWDXNT-IXOXFDKPSA-N 0.000 description 12
- YUJLIIRMIAGMCQ-CIUDSAMLSA-N Ser-Leu-Ser Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(O)=O YUJLIIRMIAGMCQ-CIUDSAMLSA-N 0.000 description 12
- DJEVQCWNMQOABE-RCOVLWMOSA-N Val-Gly-Asp Chemical compound CC(C)[C@@H](C(=O)NCC(=O)N[C@@H](CC(=O)O)C(=O)O)N DJEVQCWNMQOABE-RCOVLWMOSA-N 0.000 description 12
- 229960002297 fenofibrate Drugs 0.000 description 12
- YMTINGFKWWXKFG-UHFFFAOYSA-N fenofibrate Chemical compound C1=CC(OC(C)(C)C(=O)OC(C)C)=CC=C1C(=O)C1=CC=C(Cl)C=C1 YMTINGFKWWXKFG-UHFFFAOYSA-N 0.000 description 12
- 102000004196 processed proteins & peptides Human genes 0.000 description 12
- JBGSZRYCXBPWGX-BQBZGAKWSA-N Ala-Arg-Gly Chemical compound OC(=O)CNC(=O)[C@@H](NC(=O)[C@@H](N)C)CCCN=C(N)N JBGSZRYCXBPWGX-BQBZGAKWSA-N 0.000 description 11
- LECUEEHKUFYOOV-ZJDVBMNYSA-N Thr-Thr-Val Chemical compound CC(C)[C@@H](C(O)=O)NC(=O)[C@H]([C@@H](C)O)NC(=O)[C@@H](N)[C@@H](C)O LECUEEHKUFYOOV-ZJDVBMNYSA-N 0.000 description 11
- 230000035772 mutation Effects 0.000 description 11
- 125000003729 nucleotide group Chemical group 0.000 description 11
- VKKYFICVTYKFIO-CIUDSAMLSA-N Arg-Ala-Glu Chemical compound OC(=O)CC[C@@H](C(O)=O)NC(=O)[C@H](C)NC(=O)[C@@H](N)CCCN=C(N)N VKKYFICVTYKFIO-CIUDSAMLSA-N 0.000 description 10
- KWKQGHSSNHPGOW-BQBZGAKWSA-N Arg-Ala-Gly Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](C)C(=O)NCC(O)=O KWKQGHSSNHPGOW-BQBZGAKWSA-N 0.000 description 10
- 241000282693 Cercopithecidae Species 0.000 description 10
- CAODKDAPYGUMLK-FXQIFTODSA-N Met-Asn-Ser Chemical compound CSCC[C@H](N)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CO)C(O)=O CAODKDAPYGUMLK-FXQIFTODSA-N 0.000 description 10
- FIDMVVBUOCMMJG-CIUDSAMLSA-N Ser-Asn-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@@H](N)CO FIDMVVBUOCMMJG-CIUDSAMLSA-N 0.000 description 10
- IOVHBRCQOGWAQH-ZKWXMUAHSA-N Ser-Gly-Ile Chemical compound [H]N[C@@H](CO)C(=O)NCC(=O)N[C@@H]([C@@H](C)CC)C(O)=O IOVHBRCQOGWAQH-ZKWXMUAHSA-N 0.000 description 10
- 108010008685 alanyl-glutamyl-aspartic acid Proteins 0.000 description 10
- 125000000539 amino acid group Chemical group 0.000 description 10
- 238000001727 in vivo Methods 0.000 description 10
- 239000002773 nucleotide Substances 0.000 description 10
- 229920001184 polypeptide Polymers 0.000 description 10
- 230000009467 reduction Effects 0.000 description 10
- SXFPZRRVWSUYII-KBIXCLLPSA-N Gln-Ser-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H](CO)NC(=O)[C@H](CCC(=O)N)N SXFPZRRVWSUYII-KBIXCLLPSA-N 0.000 description 9
- OACQOWPRWGNKTP-AVGNSLFASA-N Gln-Tyr-Asp Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(O)=O)C(O)=O OACQOWPRWGNKTP-AVGNSLFASA-N 0.000 description 9
- KSOBNUBCYHGUKH-UWVGGRQHSA-N Gly-Val-Val Chemical compound CC(C)[C@@H](C(O)=O)NC(=O)[C@H](C(C)C)NC(=O)CN KSOBNUBCYHGUKH-UWVGGRQHSA-N 0.000 description 9
- 108010010234 HDL Lipoproteins Proteins 0.000 description 9
- 102000015779 HDL Lipoproteins Human genes 0.000 description 9
- -1 His Chemical compound 0.000 description 9
- DFFTXLCCDFYRKD-MBLNEYKQSA-N Ile-Gly-Thr Chemical compound CC[C@H](C)[C@@H](C(=O)NCC(=O)N[C@@H]([C@@H](C)O)C(=O)O)N DFFTXLCCDFYRKD-MBLNEYKQSA-N 0.000 description 9
- QESXLSQLQHHTIX-RHYQMDGZSA-N Leu-Val-Thr Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O QESXLSQLQHHTIX-RHYQMDGZSA-N 0.000 description 9
- KZNQNBZMBZJQJO-UHFFFAOYSA-N N-glycyl-L-proline Natural products NCC(=O)N1CCCC1C(O)=O KZNQNBZMBZJQJO-UHFFFAOYSA-N 0.000 description 9
- XALFIVXGQUEGKV-JSGCOSHPSA-N Phe-Val-Gly Chemical compound OC(=O)CNC(=O)[C@H](C(C)C)NC(=O)[C@@H](N)CC1=CC=CC=C1 XALFIVXGQUEGKV-JSGCOSHPSA-N 0.000 description 9
- KIEIJCFVGZCUAS-MELADBBJSA-N Ser-Tyr-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CC2=CC=C(C=C2)O)NC(=O)[C@H](CO)N)C(=O)O KIEIJCFVGZCUAS-MELADBBJSA-N 0.000 description 9
- LMKKMCGTDANZTR-BZSNNMDCSA-N Tyr-Phe-Asp Chemical compound C([C@H](N)C(=O)N[C@@H](CC=1C=CC=CC=1)C(=O)N[C@@H](CC(O)=O)C(O)=O)C1=CC=C(O)C=C1 LMKKMCGTDANZTR-BZSNNMDCSA-N 0.000 description 9
- 230000004071 biological effect Effects 0.000 description 9
- 201000011510 cancer Diseases 0.000 description 9
- 210000004027 cell Anatomy 0.000 description 9
- 108010077515 glycylproline Proteins 0.000 description 9
- 108010070409 phenylalanyl-glycyl-glycine Proteins 0.000 description 9
- 238000002198 surface plasmon resonance spectroscopy Methods 0.000 description 9
- 108010038745 tryptophylglycine Proteins 0.000 description 9
- JSHWXQIZOCVWIA-ZKWXMUAHSA-N Asp-Ser-Val Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(O)=O JSHWXQIZOCVWIA-ZKWXMUAHSA-N 0.000 description 8
- ZZLDMBMFKZFQMU-NRPADANISA-N Gln-Val-Ala Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](C)C(O)=O ZZLDMBMFKZFQMU-NRPADANISA-N 0.000 description 8
- NVTPVQLIZCOJFK-FOHZUACHSA-N Gly-Thr-Asp Chemical compound [H]NCC(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(O)=O)C(O)=O NVTPVQLIZCOJFK-FOHZUACHSA-N 0.000 description 8
- TVTZEOHWHUVYCG-KYNKHSRBSA-N Gly-Thr-Thr Chemical compound [H]NCC(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O TVTZEOHWHUVYCG-KYNKHSRBSA-N 0.000 description 8
- CKLJMWTZIZZHCS-REOHCLBHSA-N L-aspartic acid Chemical compound OC(=O)[C@@H](N)CC(O)=O CKLJMWTZIZZHCS-REOHCLBHSA-N 0.000 description 8
- 108010007622 LDL Lipoproteins Proteins 0.000 description 8
- 102000007330 LDL Lipoproteins Human genes 0.000 description 8
- LCNASHSOFMRYFO-WDCWCFNPSA-N Leu-Thr-Gln Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@H](C(O)=O)CCC(N)=O LCNASHSOFMRYFO-WDCWCFNPSA-N 0.000 description 8
- YIRIDPUGZKHMHT-ACRUOGEOSA-N Leu-Tyr-Tyr Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O YIRIDPUGZKHMHT-ACRUOGEOSA-N 0.000 description 8
- MVJRBCJCRYGCKV-GVXVVHGQSA-N Leu-Val-Gln Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(N)=O)C(O)=O MVJRBCJCRYGCKV-GVXVVHGQSA-N 0.000 description 8
- UWKNTTJNVSYXPC-CIUDSAMLSA-N Lys-Ala-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@H](C)NC(=O)[C@@H](N)CCCCN UWKNTTJNVSYXPC-CIUDSAMLSA-N 0.000 description 8
- ZENDEDYRYVHBEG-SRVKXCTJSA-N Phe-Asp-Asp Chemical compound OC(=O)C[C@@H](C(O)=O)NC(=O)[C@H](CC(O)=O)NC(=O)[C@@H](N)CC1=CC=CC=C1 ZENDEDYRYVHBEG-SRVKXCTJSA-N 0.000 description 8
- AFXCXDQNRXTSBD-FJXKBIBVSA-N Pro-Gly-Thr Chemical compound [H]N1CCC[C@H]1C(=O)NCC(=O)N[C@@H]([C@@H](C)O)C(O)=O AFXCXDQNRXTSBD-FJXKBIBVSA-N 0.000 description 8
- 239000002246 antineoplastic agent Substances 0.000 description 8
- 229940127089 cytotoxic agent Drugs 0.000 description 8
- 108010082286 glycyl-seryl-alanine Proteins 0.000 description 8
- 230000004048 modification Effects 0.000 description 8
- 238000012986 modification Methods 0.000 description 8
- ZDILXFDENZVOTL-BPNCWPANSA-N Ala-Val-Tyr Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O ZDILXFDENZVOTL-BPNCWPANSA-N 0.000 description 7
- ZBYLEBZCVKLPCY-FXQIFTODSA-N Asp-Ser-Arg Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O ZBYLEBZCVKLPCY-FXQIFTODSA-N 0.000 description 7
- 241000283707 Capra Species 0.000 description 7
- 238000002965 ELISA Methods 0.000 description 7
- UQJNXZSSGQIPIQ-FBCQKBJTSA-N Gly-Gly-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)CNC(=O)CN UQJNXZSSGQIPIQ-FBCQKBJTSA-N 0.000 description 7
- RVGMVLVBDRQVKB-UWVGGRQHSA-N Gly-Met-His Chemical compound CSCC[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)NC(=O)CN RVGMVLVBDRQVKB-UWVGGRQHSA-N 0.000 description 7
- 108010001336 Horseradish Peroxidase Proteins 0.000 description 7
- DCXYFEDJOCDNAF-REOHCLBHSA-N L-asparagine Chemical compound OC(=O)[C@@H](N)CC(N)=O DCXYFEDJOCDNAF-REOHCLBHSA-N 0.000 description 7
- OUYCCCASQSFEME-QMMMGPOBSA-N L-tyrosine Chemical compound OC(=O)[C@@H](N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-QMMMGPOBSA-N 0.000 description 7
- QLFAPXUXEBAWEK-NHCYSSNCSA-N Lys-Val-Asp Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CC(O)=O)C(O)=O QLFAPXUXEBAWEK-NHCYSSNCSA-N 0.000 description 7
- 101100001708 Mus musculus Angptl4 gene Proteins 0.000 description 7
- 210000004899 c-terminal region Anatomy 0.000 description 7
- 231100000599 cytotoxic agent Toxicity 0.000 description 7
- 238000010494 dissociation reaction Methods 0.000 description 7
- 230000005593 dissociations Effects 0.000 description 7
- 238000002474 experimental method Methods 0.000 description 7
- 235000021588 free fatty acids Nutrition 0.000 description 7
- 108010050848 glycylleucine Proteins 0.000 description 7
- 150000005830 nonesterified fatty acids Chemical class 0.000 description 7
- 238000012360 testing method Methods 0.000 description 7
- 230000001225 therapeutic effect Effects 0.000 description 7
- MAEQBGQTDWDSJQ-LSJOCFKGSA-N Ala-Met-His Chemical compound C[C@@H](C(=O)N[C@@H](CCSC)C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)N MAEQBGQTDWDSJQ-LSJOCFKGSA-N 0.000 description 6
- 101710085848 Angiopoietin-related protein 3 Proteins 0.000 description 6
- 102100025668 Angiopoietin-related protein 3 Human genes 0.000 description 6
- SLKLLQWZQHXYSV-CIUDSAMLSA-N Asn-Ala-Lys Chemical compound NC(=O)C[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@@H](CCCCN)C(O)=O SLKLLQWZQHXYSV-CIUDSAMLSA-N 0.000 description 6
- RTFXPCYMDYBZNQ-SRVKXCTJSA-N Asn-Tyr-Asn Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(N)=O)C(O)=O RTFXPCYMDYBZNQ-SRVKXCTJSA-N 0.000 description 6
- LRCIOEVFVGXZKB-BZSNNMDCSA-N Asn-Tyr-Tyr Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O LRCIOEVFVGXZKB-BZSNNMDCSA-N 0.000 description 6
- KOWYNSKRPUWSFG-IHPCNDPISA-N Asp-Phe-Trp Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CC2=CNC3=CC=CC=C32)C(=O)O)NC(=O)[C@H](CC(=O)O)N KOWYNSKRPUWSFG-IHPCNDPISA-N 0.000 description 6
- AVZHGSCDKIQZPQ-CIUDSAMLSA-N Glu-Arg-Ala Chemical compound C[C@H](NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@@H](N)CCC(O)=O)C(O)=O AVZHGSCDKIQZPQ-CIUDSAMLSA-N 0.000 description 6
- IRJWAYCXIYUHQE-WHFBIAKZSA-N Gly-Ser-Ala Chemical compound OC(=O)[C@H](C)NC(=O)[C@H](CO)NC(=O)CN IRJWAYCXIYUHQE-WHFBIAKZSA-N 0.000 description 6
- 108010021625 Immunoglobulin Fragments Proteins 0.000 description 6
- 102000008394 Immunoglobulin Fragments Human genes 0.000 description 6
- COLNVLDHVKWLRT-QMMMGPOBSA-N L-phenylalanine Chemical compound OC(=O)[C@@H](N)CC1=CC=CC=C1 COLNVLDHVKWLRT-QMMMGPOBSA-N 0.000 description 6
- AYFVYJQAPQTCCC-GBXIJSLDSA-N L-threonine Chemical compound C[C@@H](O)[C@H](N)C(O)=O AYFVYJQAPQTCCC-GBXIJSLDSA-N 0.000 description 6
- LZDNBBYBDGBADK-UHFFFAOYSA-N L-valyl-L-tryptophan Natural products C1=CC=C2C(CC(NC(=O)C(N)C(C)C)C(O)=O)=CNC2=C1 LZDNBBYBDGBADK-UHFFFAOYSA-N 0.000 description 6
- HPBCTWSUJOGJSH-MNXVOIDGSA-N Leu-Glu-Ile Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O HPBCTWSUJOGJSH-MNXVOIDGSA-N 0.000 description 6
- YYKZDTVQHTUKDW-RYUDHWBXSA-N Phe-Gly-Gln Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)NCC(=O)N[C@@H](CCC(=O)N)C(=O)O)N YYKZDTVQHTUKDW-RYUDHWBXSA-N 0.000 description 6
- VZQRNAYURWAEFE-KKUMJFAQSA-N Ser-Leu-Phe Chemical compound OC[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 VZQRNAYURWAEFE-KKUMJFAQSA-N 0.000 description 6
- NADLKBTYNKUJEP-KATARQTJSA-N Ser-Thr-Leu Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(C)C)C(O)=O NADLKBTYNKUJEP-KATARQTJSA-N 0.000 description 6
- AXKJPUBALUNJEO-UBHSHLNASA-N Ser-Trp-Asn Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(=O)N[C@@H](CC(N)=O)C(O)=O AXKJPUBALUNJEO-UBHSHLNASA-N 0.000 description 6
- ZVBCMFDJIMUELU-BZSNNMDCSA-N Ser-Tyr-Phe Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)O)NC(=O)[C@H](CC2=CC=C(C=C2)O)NC(=O)[C@H](CO)N ZVBCMFDJIMUELU-BZSNNMDCSA-N 0.000 description 6
- SVGAWGVHFIYAEE-JSGCOSHPSA-N Trp-Gly-Gln Chemical compound C1=CC=C2C(C[C@H](N)C(=O)NCC(=O)N[C@@H](CCC(N)=O)C(O)=O)=CNC2=C1 SVGAWGVHFIYAEE-JSGCOSHPSA-N 0.000 description 6
- JWHOIHCOHMZSAR-QWRGUYRKSA-N Tyr-Asp-Gly Chemical compound OC(=O)CNC(=O)[C@H](CC(O)=O)NC(=O)[C@@H](N)CC1=CC=C(O)C=C1 JWHOIHCOHMZSAR-QWRGUYRKSA-N 0.000 description 6
- WPVGRKLNHJJCEN-BZSNNMDCSA-N Tyr-Asp-Phe Chemical compound C([C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC=1C=CC=CC=1)C(O)=O)C1=CC=C(O)C=C1 WPVGRKLNHJJCEN-BZSNNMDCSA-N 0.000 description 6
- RWOKVQUCENPXGE-IHRRRGAJSA-N Tyr-Ser-Arg Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CO)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O RWOKVQUCENPXGE-IHRRRGAJSA-N 0.000 description 6
- AZSHAZJLOZQYAY-FXQIFTODSA-N Val-Ala-Ser Chemical compound CC(C)[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@@H](CO)C(O)=O AZSHAZJLOZQYAY-FXQIFTODSA-N 0.000 description 6
- NZYNRRGJJVSSTJ-GUBZILKMSA-N Val-Ser-Val Chemical compound CC(C)[C@H](N)C(=O)N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(O)=O NZYNRRGJJVSSTJ-GUBZILKMSA-N 0.000 description 6
- HTONZBWRYUKUKC-RCWTZXSCSA-N Val-Thr-Val Chemical compound CC(C)[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C(C)C)C(O)=O HTONZBWRYUKUKC-RCWTZXSCSA-N 0.000 description 6
- 238000003556 assay Methods 0.000 description 6
- 239000002552 dosage form Substances 0.000 description 6
- 230000029142 excretion Effects 0.000 description 6
- 108010063718 gamma-glutamylaspartic acid Proteins 0.000 description 6
- 238000001990 intravenous administration Methods 0.000 description 6
- 108010053725 prolylvaline Proteins 0.000 description 6
- 241000894007 species Species 0.000 description 6
- 239000003981 vehicle Substances 0.000 description 6
- GNMQDOGFWYWPNM-LAEOZQHASA-N Gln-Gly-Ile Chemical compound CC[C@H](C)[C@H](NC(=O)CNC(=O)[C@@H](N)CCC(N)=O)C(O)=O GNMQDOGFWYWPNM-LAEOZQHASA-N 0.000 description 5
- INGJLBQKTRJLFO-UKJIMTQDSA-N Glu-Ile-Val Chemical compound CC(C)[C@@H](C(O)=O)NC(=O)[C@H]([C@@H](C)CC)NC(=O)[C@@H](N)CCC(O)=O INGJLBQKTRJLFO-UKJIMTQDSA-N 0.000 description 5
- 241000282412 Homo Species 0.000 description 5
- NZGTYCMLUGYMCV-XUXIUFHCSA-N Ile-Lys-Arg Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCCN=C(N)N)C(=O)O)N NZGTYCMLUGYMCV-XUXIUFHCSA-N 0.000 description 5
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 5
- 108010028554 LDL Cholesterol Proteins 0.000 description 5
- 241000699660 Mus musculus Species 0.000 description 5
- YCCUXNNKXDGMAM-KKUMJFAQSA-N Phe-Leu-Ser Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(O)=O YCCUXNNKXDGMAM-KKUMJFAQSA-N 0.000 description 5
- HDBOEVPDIDDEPC-CIUDSAMLSA-N Ser-Lys-Asn Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(N)=O)C(O)=O HDBOEVPDIDDEPC-CIUDSAMLSA-N 0.000 description 5
- SCCKSNREWHMKOJ-SRVKXCTJSA-N Tyr-Asn-Ser Chemical compound N[C@@H](Cc1ccc(O)cc1)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CO)C(O)=O SCCKSNREWHMKOJ-SRVKXCTJSA-N 0.000 description 5
- WTTRJMAZPDHPGS-KKXDTOCCSA-N Tyr-Phe-Ala Chemical compound C[C@H](NC(=O)[C@H](Cc1ccccc1)NC(=O)[C@@H](N)Cc1ccc(O)cc1)C(O)=O WTTRJMAZPDHPGS-KKXDTOCCSA-N 0.000 description 5
- 230000009102 absorption Effects 0.000 description 5
- 238000010521 absorption reaction Methods 0.000 description 5
- 108010041407 alanylaspartic acid Proteins 0.000 description 5
- 210000004369 blood Anatomy 0.000 description 5
- 239000008280 blood Substances 0.000 description 5
- 230000008859 change Effects 0.000 description 5
- 150000001875 compounds Chemical group 0.000 description 5
- 239000002254 cytotoxic agent Substances 0.000 description 5
- 238000012217 deletion Methods 0.000 description 5
- 230000037430 deletion Effects 0.000 description 5
- 229940079593 drug Drugs 0.000 description 5
- 235000013305 food Nutrition 0.000 description 5
- 108010078144 glutaminyl-glycine Proteins 0.000 description 5
- 238000001802 infusion Methods 0.000 description 5
- 108010034529 leucyl-lysine Proteins 0.000 description 5
- 210000004897 n-terminal region Anatomy 0.000 description 5
- 238000007920 subcutaneous administration Methods 0.000 description 5
- 238000010254 subcutaneous injection Methods 0.000 description 5
- 239000007929 subcutaneous injection Substances 0.000 description 5
- 238000002560 therapeutic procedure Methods 0.000 description 5
- 238000011830 transgenic mouse model Methods 0.000 description 5
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N (+)-Biotin Chemical compound N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 description 4
- NFGXHKASABOEEW-UHFFFAOYSA-N 1-methylethyl 11-methoxy-3,7,11-trimethyl-2,4-dodecadienoate Chemical compound COC(C)(C)CCCC(C)CC=CC(C)=CC(=O)OC(C)C NFGXHKASABOEEW-UHFFFAOYSA-N 0.000 description 4
- MFMDKJIPHSWSBM-GUBZILKMSA-N Ala-Lys-Glu Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCC(O)=O)C(O)=O MFMDKJIPHSWSBM-GUBZILKMSA-N 0.000 description 4
- NINQYGGNRIBFSC-CIUDSAMLSA-N Ala-Lys-Ser Chemical compound NCCCC[C@H](NC(=O)[C@@H](N)C)C(=O)N[C@@H](CO)C(O)=O NINQYGGNRIBFSC-CIUDSAMLSA-N 0.000 description 4
- PQWTZSNVWSOFFK-FXQIFTODSA-N Arg-Asp-Asn Chemical compound C(C[C@@H](C(=O)N[C@@H](CC(=O)O)C(=O)N[C@@H](CC(=O)N)C(=O)O)N)CN=C(N)N PQWTZSNVWSOFFK-FXQIFTODSA-N 0.000 description 4
- VLIJAPRTSXSGFY-STQMWFEESA-N Arg-Tyr-Gly Chemical compound NC(N)=NCCC[C@H](N)C(=O)N[C@H](C(=O)NCC(O)=O)CC1=CC=C(O)C=C1 VLIJAPRTSXSGFY-STQMWFEESA-N 0.000 description 4
- ULBHWNVWSCJLCO-NHCYSSNCSA-N Arg-Val-Glu Chemical compound OC(=O)CC[C@@H](C(O)=O)NC(=O)[C@H](C(C)C)NC(=O)[C@@H](N)CCCN=C(N)N ULBHWNVWSCJLCO-NHCYSSNCSA-N 0.000 description 4
- GLWFAWNYGWBMOC-SRVKXCTJSA-N Asn-Leu-Leu Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC(C)C)C(O)=O GLWFAWNYGWBMOC-SRVKXCTJSA-N 0.000 description 4
- RBOBTTLFPRSXKZ-BZSNNMDCSA-N Asn-Phe-Tyr Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O RBOBTTLFPRSXKZ-BZSNNMDCSA-N 0.000 description 4
- CPYHLXSGDBDULY-IHPCNDPISA-N Asn-Trp-Phe Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O CPYHLXSGDBDULY-IHPCNDPISA-N 0.000 description 4
- XPGVTUBABLRGHY-BIIVOSGPSA-N Asp-Ala-Pro Chemical compound C[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CC(=O)O)N XPGVTUBABLRGHY-BIIVOSGPSA-N 0.000 description 4
- 102100035360 Cerebellar degeneration-related antigen 1 Human genes 0.000 description 4
- YFKWIIRWHGKSQQ-WFBYXXMGSA-N Cys-Trp-Ala Chemical compound C[C@@H](C(=O)O)NC(=O)[C@H](CC1=CNC2=CC=CC=C21)NC(=O)[C@H](CS)N YFKWIIRWHGKSQQ-WFBYXXMGSA-N 0.000 description 4
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 4
- RDDSZZJOKDVPAE-ACZMJKKPSA-N Glu-Asn-Ser Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CO)C(O)=O RDDSZZJOKDVPAE-ACZMJKKPSA-N 0.000 description 4
- OAGVHWYIBZMWLA-YFKPBYRVSA-N Glu-Gly-Gly Chemical compound OC(=O)CC[C@H](N)C(=O)NCC(=O)NCC(O)=O OAGVHWYIBZMWLA-YFKPBYRVSA-N 0.000 description 4
- JZJGEKDPWVJOLD-QEWYBTABSA-N Glu-Phe-Ile Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O JZJGEKDPWVJOLD-QEWYBTABSA-N 0.000 description 4
- PMSDOVISAARGAV-FHWLQOOXSA-N Glu-Tyr-Phe Chemical compound C([C@H](NC(=O)[C@H](CCC(O)=O)N)C(=O)N[C@@H](CC=1C=CC=CC=1)C(O)=O)C1=CC=C(O)C=C1 PMSDOVISAARGAV-FHWLQOOXSA-N 0.000 description 4
- IWAXHBCACVWNHT-BQBZGAKWSA-N Gly-Asp-Arg Chemical compound NCC(=O)N[C@@H](CC(O)=O)C(=O)N[C@H](C(O)=O)CCCN=C(N)N IWAXHBCACVWNHT-BQBZGAKWSA-N 0.000 description 4
- LOEANKRDMMVOGZ-YUMQZZPRSA-N Gly-Lys-Asp Chemical compound NCCCC[C@H](NC(=O)CN)C(=O)N[C@@H](CC(O)=O)C(O)=O LOEANKRDMMVOGZ-YUMQZZPRSA-N 0.000 description 4
- JBCLFWXMTIKCCB-UHFFFAOYSA-N H-Gly-Phe-OH Natural products NCC(=O)NC(C(O)=O)CC1=CC=CC=C1 JBCLFWXMTIKCCB-UHFFFAOYSA-N 0.000 description 4
- 108010093488 His-His-His-His-His-His Proteins 0.000 description 4
- MKWFGXSFLYNTKC-XIRDDKMYSA-N His-Trp-Asp Chemical compound C1=CC=C2C(=C1)C(=CN2)C[C@@H](C(=O)N[C@@H](CC(=O)O)C(=O)O)NC(=O)[C@H](CC3=CN=CN3)N MKWFGXSFLYNTKC-XIRDDKMYSA-N 0.000 description 4
- 101000737793 Homo sapiens Cerebellar degeneration-related antigen 1 Proteins 0.000 description 4
- 108060003951 Immunoglobulin Proteins 0.000 description 4
- 108010054477 Immunoglobulin Fab Fragments Proteins 0.000 description 4
- 102000001706 Immunoglobulin Fab Fragments Human genes 0.000 description 4
- ZDXPYRJPNDTMRX-VKHMYHEASA-N L-glutamine Chemical compound OC(=O)[C@@H](N)CCC(N)=O ZDXPYRJPNDTMRX-VKHMYHEASA-N 0.000 description 4
- KDXKERNSBIXSRK-YFKPBYRVSA-N L-lysine Chemical compound NCCCC[C@H](N)C(O)=O KDXKERNSBIXSRK-YFKPBYRVSA-N 0.000 description 4
- QCSFMCFHVGTLFF-NHCYSSNCSA-N Leu-Asp-Val Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](C(C)C)C(O)=O QCSFMCFHVGTLFF-NHCYSSNCSA-N 0.000 description 4
- JRJLGNFWYFSJHB-HOCLYGCPSA-N Leu-Gly-Trp Chemical compound [H]N[C@@H](CC(C)C)C(=O)NCC(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(O)=O JRJLGNFWYFSJHB-HOCLYGCPSA-N 0.000 description 4
- FAELBUXXFQLUAX-AJNGGQMLSA-N Leu-Leu-Ile Chemical compound CC[C@H](C)[C@@H](C(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@@H](N)CC(C)C FAELBUXXFQLUAX-AJNGGQMLSA-N 0.000 description 4
- SIGZKCWZEBFNAK-QAETUUGQSA-N Leu-Ser-Ser-Tyr Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H](CO)C(=O)N[C@@H](CO)C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 SIGZKCWZEBFNAK-QAETUUGQSA-N 0.000 description 4
- YNNPKXBBRZVIRX-IHRRRGAJSA-N Lys-Arg-Leu Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(C)C)C(O)=O YNNPKXBBRZVIRX-IHRRRGAJSA-N 0.000 description 4
- ZXFRGTAIIZHNHG-AJNGGQMLSA-N Lys-Ile-Leu Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC(C)C)C(=O)O)NC(=O)[C@H](CCCCN)N ZXFRGTAIIZHNHG-AJNGGQMLSA-N 0.000 description 4
- FVKRQMQQFGBXHV-QXEWZRGKSA-N Met-Asp-Val Chemical compound CSCC[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](C(C)C)C(O)=O FVKRQMQQFGBXHV-QXEWZRGKSA-N 0.000 description 4
- WUGMRIBZSVSJNP-UHFFFAOYSA-N N-L-alanyl-L-tryptophan Natural products C1=CC=C2C(CC(NC(=O)C(N)C)C(O)=O)=CNC2=C1 WUGMRIBZSVSJNP-UHFFFAOYSA-N 0.000 description 4
- JEGFCFLCRSJCMA-IHRRRGAJSA-N Phe-Arg-Ser Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CO)C(=O)O)N JEGFCFLCRSJCMA-IHRRRGAJSA-N 0.000 description 4
- WEDZFLRYSIDIRX-IHRRRGAJSA-N Phe-Ser-Arg Chemical compound NC(=N)NCCC[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CC1=CC=CC=C1 WEDZFLRYSIDIRX-IHRRRGAJSA-N 0.000 description 4
- YMIZSYUAZJSOFL-SRVKXCTJSA-N Phe-Ser-Asn Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(N)=O)C(O)=O YMIZSYUAZJSOFL-SRVKXCTJSA-N 0.000 description 4
- KIQUCMUULDXTAZ-HJOGWXRNSA-N Phe-Tyr-Tyr Chemical compound N[C@@H](Cc1ccccc1)C(=O)N[C@@H](Cc1ccc(O)cc1)C(=O)N[C@@H](Cc1ccc(O)cc1)C(O)=O KIQUCMUULDXTAZ-HJOGWXRNSA-N 0.000 description 4
- DYJTXTCEXMCPBF-UFYCRDLUSA-N Pro-Tyr-Phe Chemical compound C1C[C@H](NC1)C(=O)N[C@@H](CC2=CC=C(C=C2)O)C(=O)N[C@@H](CC3=CC=CC=C3)C(=O)O DYJTXTCEXMCPBF-UFYCRDLUSA-N 0.000 description 4
- HZWAHWQZPSXNCB-BPUTZDHNSA-N Ser-Arg-Trp Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(O)=O HZWAHWQZPSXNCB-BPUTZDHNSA-N 0.000 description 4
- ICHZYBVODUVUKN-SRVKXCTJSA-N Ser-Asn-Tyr Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O ICHZYBVODUVUKN-SRVKXCTJSA-N 0.000 description 4
- IXCHOHLPHNGFTJ-YUMQZZPRSA-N Ser-Gly-His Chemical compound C1=C(NC=N1)C[C@@H](C(=O)O)NC(=O)CNC(=O)[C@H](CO)N IXCHOHLPHNGFTJ-YUMQZZPRSA-N 0.000 description 4
- SRSPTFBENMJHMR-WHFBIAKZSA-N Ser-Ser-Gly Chemical compound OC[C@H](N)C(=O)N[C@@H](CO)C(=O)NCC(O)=O SRSPTFBENMJHMR-WHFBIAKZSA-N 0.000 description 4
- JGUWRQWULDWNCM-FXQIFTODSA-N Ser-Val-Ser Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CO)C(O)=O JGUWRQWULDWNCM-FXQIFTODSA-N 0.000 description 4
- CTDPLKMBVALCGN-JSGCOSHPSA-N Tyr-Gly-Val Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)NCC(=O)N[C@@H](C(C)C)C(O)=O CTDPLKMBVALCGN-JSGCOSHPSA-N 0.000 description 4
- RMRFSFXLFWWAJZ-HJOGWXRNSA-N Tyr-Tyr-Tyr Chemical compound C([C@H](N)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(O)=O)C1=CC=C(O)C=C1 RMRFSFXLFWWAJZ-HJOGWXRNSA-N 0.000 description 4
- 108010062497 VLDL Lipoproteins Proteins 0.000 description 4
- RWOGENDAOGMHLX-DCAQKATOSA-N Val-Lys-Ala Chemical compound C[C@@H](C(=O)O)NC(=O)[C@H](CCCCN)NC(=O)[C@H](C(C)C)N RWOGENDAOGMHLX-DCAQKATOSA-N 0.000 description 4
- PGBMPFKFKXYROZ-UFYCRDLUSA-N Val-Tyr-Phe Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)N[C@@H](CC2=CC=CC=C2)C(=O)O)N PGBMPFKFKXYROZ-UFYCRDLUSA-N 0.000 description 4
- 102000005789 Vascular Endothelial Growth Factors Human genes 0.000 description 4
- 108010019530 Vascular Endothelial Growth Factors Proteins 0.000 description 4
- 108010081404 acein-2 Proteins 0.000 description 4
- 108010087924 alanylproline Proteins 0.000 description 4
- 102000025171 antigen binding proteins Human genes 0.000 description 4
- 108091000831 antigen binding proteins Proteins 0.000 description 4
- 108010069926 arginyl-glycyl-serine Proteins 0.000 description 4
- 108010047857 aspartylglycine Proteins 0.000 description 4
- 210000003719 b-lymphocyte Anatomy 0.000 description 4
- 238000013270 controlled release Methods 0.000 description 4
- 230000003247 decreasing effect Effects 0.000 description 4
- 108700041286 delta Proteins 0.000 description 4
- FSXRLASFHBWESK-UHFFFAOYSA-N dipeptide phenylalanyl-tyrosine Natural products C=1C=C(O)C=CC=1CC(C(O)=O)NC(=O)C(N)CC1=CC=CC=C1 FSXRLASFHBWESK-UHFFFAOYSA-N 0.000 description 4
- 238000009472 formulation Methods 0.000 description 4
- 108010006664 gamma-glutamyl-glycyl-glycine Proteins 0.000 description 4
- 230000014509 gene expression Effects 0.000 description 4
- 108010081551 glycylphenylalanine Proteins 0.000 description 4
- 108010092114 histidylphenylalanine Proteins 0.000 description 4
- 210000004408 hybridoma Anatomy 0.000 description 4
- 229940127121 immunoconjugate Drugs 0.000 description 4
- 102000018358 immunoglobulin Human genes 0.000 description 4
- 108010083708 leucyl-aspartyl-valine Proteins 0.000 description 4
- 230000000670 limiting effect Effects 0.000 description 4
- 229940021182 non-steroidal anti-inflammatory drug Drugs 0.000 description 4
- 108010051242 phenylalanylserine Proteins 0.000 description 4
- 210000002381 plasma Anatomy 0.000 description 4
- 238000002360 preparation method Methods 0.000 description 4
- 239000002904 solvent Substances 0.000 description 4
- 230000009870 specific binding Effects 0.000 description 4
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 description 3
- BTYTYHBSJKQBQA-GCJQMDKQSA-N Ala-Asp-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CC(=O)O)NC(=O)[C@H](C)N)O BTYTYHBSJKQBQA-GCJQMDKQSA-N 0.000 description 3
- GSHKMNKPMLXSQW-KBIXCLLPSA-N Ala-Ile-Gln Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)O)NC(=O)[C@H](C)N GSHKMNKPMLXSQW-KBIXCLLPSA-N 0.000 description 3
- DHBKYZYFEXXUAK-ONGXEEELSA-N Ala-Phe-Gly Chemical compound OC(=O)CNC(=O)[C@@H](NC(=O)[C@@H](N)C)CC1=CC=CC=C1 DHBKYZYFEXXUAK-ONGXEEELSA-N 0.000 description 3
- WNHNMKOFKCHKKD-BFHQHQDPSA-N Ala-Thr-Gly Chemical compound [H]N[C@@H](C)C(=O)N[C@@H]([C@@H](C)O)C(=O)NCC(O)=O WNHNMKOFKCHKKD-BFHQHQDPSA-N 0.000 description 3
- 102000013918 Apolipoproteins E Human genes 0.000 description 3
- 108010025628 Apolipoproteins E Proteins 0.000 description 3
- OTOXOKCIIQLMFH-KZVJFYERSA-N Arg-Ala-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)[C@H](C)NC(=O)[C@@H](N)CCCN=C(N)N OTOXOKCIIQLMFH-KZVJFYERSA-N 0.000 description 3
- CYXCAHZVPFREJD-LURJTMIESA-N Arg-Gly-Gly Chemical compound NC(=N)NCCC[C@H](N)C(=O)NCC(=O)NCC(O)=O CYXCAHZVPFREJD-LURJTMIESA-N 0.000 description 3
- OQCWXQJLCDPRHV-UWVGGRQHSA-N Arg-Gly-Leu Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)NCC(=O)N[C@@H](CC(C)C)C(O)=O OQCWXQJLCDPRHV-UWVGGRQHSA-N 0.000 description 3
- UGZUVYDKAYNCII-ULQDDVLXSA-N Arg-Phe-Leu Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CC(C)C)C(O)=O UGZUVYDKAYNCII-ULQDDVLXSA-N 0.000 description 3
- ZKDGORKGHPCZOV-DCAQKATOSA-N Asn-His-Arg Chemical compound C1=C(NC=N1)C[C@@H](C(=O)N[C@@H](CCCN=C(N)N)C(=O)O)NC(=O)[C@H](CC(=O)N)N ZKDGORKGHPCZOV-DCAQKATOSA-N 0.000 description 3
- PNHQRQTVBRDIEF-CIUDSAMLSA-N Asn-Leu-Ala Chemical compound C[C@@H](C(=O)O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CC(=O)N)N PNHQRQTVBRDIEF-CIUDSAMLSA-N 0.000 description 3
- UPAGTDJAORYMEC-VHWLVUOQSA-N Asn-Trp-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H](CC1=CNC2=CC=CC=C21)NC(=O)[C@H](CC(=O)N)N UPAGTDJAORYMEC-VHWLVUOQSA-N 0.000 description 3
- QUCCLIXMVPIVOB-BZSNNMDCSA-N Asn-Tyr-Phe Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)O)NC(=O)[C@H](CC2=CC=C(C=C2)O)NC(=O)[C@H](CC(=O)N)N QUCCLIXMVPIVOB-BZSNNMDCSA-N 0.000 description 3
- MRQQMVZUHXUPEV-IHRRRGAJSA-N Asp-Arg-Phe Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O MRQQMVZUHXUPEV-IHRRRGAJSA-N 0.000 description 3
- USNJAPJZSGTTPX-XVSYOHENSA-N Asp-Phe-Thr Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H]([C@@H](C)O)C(O)=O USNJAPJZSGTTPX-XVSYOHENSA-N 0.000 description 3
- SXLCDCZHNCLFGZ-BPUTZDHNSA-N Asp-Pro-Trp Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(O)=O SXLCDCZHNCLFGZ-BPUTZDHNSA-N 0.000 description 3
- YUELDQUPTAYEGM-XIRDDKMYSA-N Asp-Trp-Leu Chemical compound CC(C)C[C@@H](C(=O)O)NC(=O)[C@H](CC1=CNC2=CC=CC=C21)NC(=O)[C@H](CC(=O)O)N YUELDQUPTAYEGM-XIRDDKMYSA-N 0.000 description 3
- GYNUXDMCDILYIQ-QRTARXTBSA-N Asp-Val-Trp Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CNC2=CC=CC=C21)C(=O)O)NC(=O)[C@H](CC(=O)O)N GYNUXDMCDILYIQ-QRTARXTBSA-N 0.000 description 3
- DQPOBSRQNWOBNA-GUBZILKMSA-N Gln-His-Asn Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CC(N)=O)C(O)=O DQPOBSRQNWOBNA-GUBZILKMSA-N 0.000 description 3
- VZRAXPGTUNDIDK-GUBZILKMSA-N Gln-Leu-Asn Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)O)NC(=O)[C@H](CCC(=O)N)N VZRAXPGTUNDIDK-GUBZILKMSA-N 0.000 description 3
- VUVKKXPCKILIBD-AVGNSLFASA-N Gln-Leu-His Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)NC(=O)[C@H](CCC(=O)N)N VUVKKXPCKILIBD-AVGNSLFASA-N 0.000 description 3
- XZLLTYBONVKGLO-SDDRHHMPSA-N Gln-Lys-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CCCCN)NC(=O)[C@H](CCC(=O)N)N)C(=O)O XZLLTYBONVKGLO-SDDRHHMPSA-N 0.000 description 3
- UESYBOXFJWJVSB-AVGNSLFASA-N Gln-Phe-Ser Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CO)C(O)=O UESYBOXFJWJVSB-AVGNSLFASA-N 0.000 description 3
- SGVGIVDZLSHSEN-RYUDHWBXSA-N Gln-Tyr-Gly Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)NCC(O)=O SGVGIVDZLSHSEN-RYUDHWBXSA-N 0.000 description 3
- AKDOUBMVLRCHBD-SIUGBPQLSA-N Gln-Tyr-Ile Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O AKDOUBMVLRCHBD-SIUGBPQLSA-N 0.000 description 3
- UBRQJXFDVZNYJP-AVGNSLFASA-N Gln-Tyr-Ser Chemical compound C1=CC(=CC=C1C[C@@H](C(=O)N[C@@H](CO)C(=O)O)NC(=O)[C@H](CCC(=O)N)N)O UBRQJXFDVZNYJP-AVGNSLFASA-N 0.000 description 3
- WATXSTJXNBOHKD-LAEOZQHASA-N Glu-Asp-Val Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](C(C)C)C(O)=O WATXSTJXNBOHKD-LAEOZQHASA-N 0.000 description 3
- XTZDZAXYPDISRR-MNXVOIDGSA-N Glu-Ile-Lys Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CCC(=O)O)N XTZDZAXYPDISRR-MNXVOIDGSA-N 0.000 description 3
- UCZXXMREFIETQW-AVGNSLFASA-N Glu-Tyr-Asn Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(N)=O)C(O)=O UCZXXMREFIETQW-AVGNSLFASA-N 0.000 description 3
- OCDLPQDYTJPWNG-YUMQZZPRSA-N Gly-Asn-Lys Chemical compound C(CCN)C[C@@H](C(=O)O)NC(=O)[C@H](CC(=O)N)NC(=O)CN OCDLPQDYTJPWNG-YUMQZZPRSA-N 0.000 description 3
- LURCIJSJAKFCRO-QWRGUYRKSA-N Gly-Asn-Tyr Chemical compound [H]NCC(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O LURCIJSJAKFCRO-QWRGUYRKSA-N 0.000 description 3
- LCNXZQROPKFGQK-WHFBIAKZSA-N Gly-Asp-Ser Chemical compound NCC(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CO)C(O)=O LCNXZQROPKFGQK-WHFBIAKZSA-N 0.000 description 3
- CQZDZKRHFWJXDF-WDSKDSINSA-N Gly-Gln-Ala Chemical compound OC(=O)[C@H](C)NC(=O)[C@H](CCC(N)=O)NC(=O)CN CQZDZKRHFWJXDF-WDSKDSINSA-N 0.000 description 3
- STVHDEHTKFXBJQ-LAEOZQHASA-N Gly-Glu-Ile Chemical compound [H]NCC(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O STVHDEHTKFXBJQ-LAEOZQHASA-N 0.000 description 3
- YWAQATDNEKZFFK-BYPYZUCNSA-N Gly-Gly-Ser Chemical compound NCC(=O)NCC(=O)N[C@@H](CO)C(O)=O YWAQATDNEKZFFK-BYPYZUCNSA-N 0.000 description 3
- BHPQOIPBLYJNAW-NGZCFLSTSA-N Gly-Ile-Pro Chemical compound CC[C@H](C)[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)CN BHPQOIPBLYJNAW-NGZCFLSTSA-N 0.000 description 3
- UHPAZODVFFYEEL-QWRGUYRKSA-N Gly-Leu-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)CN UHPAZODVFFYEEL-QWRGUYRKSA-N 0.000 description 3
- SOEGEPHNZOISMT-BYPYZUCNSA-N Gly-Ser-Gly Chemical compound NCC(=O)N[C@@H](CO)C(=O)NCC(O)=O SOEGEPHNZOISMT-BYPYZUCNSA-N 0.000 description 3
- WCORRBXVISTKQL-WHFBIAKZSA-N Gly-Ser-Ser Chemical compound NCC(=O)N[C@@H](CO)C(=O)N[C@@H](CO)C(O)=O WCORRBXVISTKQL-WHFBIAKZSA-N 0.000 description 3
- LCRDMSSAKLTKBU-ZDLURKLDSA-N Gly-Ser-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)CN LCRDMSSAKLTKBU-ZDLURKLDSA-N 0.000 description 3
- YXTFLTJYLIAZQG-FJXKBIBVSA-N Gly-Thr-Arg Chemical compound NCC(=O)N[C@@H]([C@H](O)C)C(=O)N[C@H](C(O)=O)CCCN=C(N)N YXTFLTJYLIAZQG-FJXKBIBVSA-N 0.000 description 3
- XHVONGZZVUUORG-WEDXCCLWSA-N Gly-Thr-Lys Chemical compound NCC(=O)N[C@@H]([C@H](O)C)C(=O)N[C@H](C(O)=O)CCCCN XHVONGZZVUUORG-WEDXCCLWSA-N 0.000 description 3
- GWCJMBNBFYBQCV-XPUUQOCRSA-N Gly-Val-Ala Chemical compound NCC(=O)N[C@@H](C(C)C)C(=O)N[C@@H](C)C(O)=O GWCJMBNBFYBQCV-XPUUQOCRSA-N 0.000 description 3
- XWUIHCZETFNRPA-IHPCNDPISA-N His-His-Trp Chemical compound C([C@H](N)C(=O)N[C@@H](CC=1NC=NC=1)C(=O)N[C@@H](CC=1C2=CC=CC=C2NC=1)C(O)=O)C1=CN=CN1 XWUIHCZETFNRPA-IHPCNDPISA-N 0.000 description 3
- BOTVMTSMOUSDRW-GMOBBJLQSA-N Ile-Arg-Asn Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CC(N)=O)C(O)=O BOTVMTSMOUSDRW-GMOBBJLQSA-N 0.000 description 3
- YKRIXHPEIZUDDY-GMOBBJLQSA-N Ile-Asn-Arg Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@H](C(O)=O)CCCN=C(N)N YKRIXHPEIZUDDY-GMOBBJLQSA-N 0.000 description 3
- GYAFMRQGWHXMII-IUKAMOBKSA-N Ile-Asp-Thr Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC(=O)O)C(=O)N[C@@H]([C@@H](C)O)C(=O)O)N GYAFMRQGWHXMII-IUKAMOBKSA-N 0.000 description 3
- SLQVFYWBGNNOTK-BYULHYEWSA-N Ile-Gly-Asn Chemical compound CC[C@H](C)[C@@H](C(=O)NCC(=O)N[C@@H](CC(=O)N)C(=O)O)N SLQVFYWBGNNOTK-BYULHYEWSA-N 0.000 description 3
- IITVUURPOYGCTD-NAKRPEOUSA-N Ile-Pro-Ala Chemical compound CC[C@H](C)[C@H](N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](C)C(O)=O IITVUURPOYGCTD-NAKRPEOUSA-N 0.000 description 3
- JODPUDMBQBIWCK-GHCJXIJMSA-N Ile-Ser-Asn Chemical compound [H]N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(N)=O)C(O)=O JODPUDMBQBIWCK-GHCJXIJMSA-N 0.000 description 3
- JZNVOBUNTWNZPW-GHCJXIJMSA-N Ile-Ser-Asp Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(=O)O)C(=O)O)N JZNVOBUNTWNZPW-GHCJXIJMSA-N 0.000 description 3
- PRTZQMBYUZFSFA-XEGUGMAKSA-N Ile-Tyr-Gly Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)NCC(=O)O)N PRTZQMBYUZFSFA-XEGUGMAKSA-N 0.000 description 3
- TYYLDKGBCJGJGW-UHFFFAOYSA-N L-tryptophan-L-tyrosine Natural products C=1NC2=CC=CC=C2C=1CC(N)C(=O)NC(C(O)=O)CC1=CC=C(O)C=C1 TYYLDKGBCJGJGW-UHFFFAOYSA-N 0.000 description 3
- 238000008214 LDL Cholesterol Methods 0.000 description 3
- SBANPBVRHYIMRR-GARJFASQSA-N Leu-Ser-Pro Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CO)C(=O)N1CCC[C@@H]1C(=O)O)N SBANPBVRHYIMRR-GARJFASQSA-N 0.000 description 3
- SBANPBVRHYIMRR-UHFFFAOYSA-N Leu-Ser-Pro Natural products CC(C)CC(N)C(=O)NC(CO)C(=O)N1CCCC1C(O)=O SBANPBVRHYIMRR-UHFFFAOYSA-N 0.000 description 3
- LINKCQUOMUDLKN-KATARQTJSA-N Leu-Thr-Cys Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CS)C(=O)O)NC(=O)[C@H](CC(C)C)N)O LINKCQUOMUDLKN-KATARQTJSA-N 0.000 description 3
- LJBVRCDPWOJOEK-PPCPHDFISA-N Leu-Thr-Ile Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O LJBVRCDPWOJOEK-PPCPHDFISA-N 0.000 description 3
- ODRREERHVHMIPT-OEAJRASXSA-N Leu-Thr-Phe Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 ODRREERHVHMIPT-OEAJRASXSA-N 0.000 description 3
- ISSAURVGLGAPDK-KKUMJFAQSA-N Leu-Tyr-Asp Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(O)=O)C(O)=O ISSAURVGLGAPDK-KKUMJFAQSA-N 0.000 description 3
- PXHCFKXNSBJSTQ-KKUMJFAQSA-N Lys-Asn-Tyr Chemical compound C1=CC(=CC=C1C[C@@H](C(=O)O)NC(=O)[C@H](CC(=O)N)NC(=O)[C@H](CCCCN)N)O PXHCFKXNSBJSTQ-KKUMJFAQSA-N 0.000 description 3
- SKRGVGLIRUGANF-AVGNSLFASA-N Lys-Leu-Glu Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(O)=O)C(O)=O SKRGVGLIRUGANF-AVGNSLFASA-N 0.000 description 3
- WRODMZBHNNPRLN-SRVKXCTJSA-N Lys-Leu-Ser Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(O)=O WRODMZBHNNPRLN-SRVKXCTJSA-N 0.000 description 3
- YTJFXEDRUOQGSP-DCAQKATOSA-N Lys-Pro-Ser Chemical compound [H]N[C@@H](CCCCN)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CO)C(O)=O YTJFXEDRUOQGSP-DCAQKATOSA-N 0.000 description 3
- IOQWIOPSKJOEKI-SRVKXCTJSA-N Lys-Ser-Leu Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(O)=O IOQWIOPSKJOEKI-SRVKXCTJSA-N 0.000 description 3
- FPQMQEOVSKMVMA-ACRUOGEOSA-N Lys-Tyr-Tyr Chemical compound C1=CC(=CC=C1C[C@@H](C(=O)N[C@@H](CC2=CC=C(C=C2)O)C(=O)O)NC(=O)[C@H](CCCCN)N)O FPQMQEOVSKMVMA-ACRUOGEOSA-N 0.000 description 3
- OZVXDDFYCQOPFD-XQQFMLRXSA-N Lys-Val-Pro Chemical compound CC(C)[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CCCCN)N OZVXDDFYCQOPFD-XQQFMLRXSA-N 0.000 description 3
- PCZOHLXUXFIOCF-UHFFFAOYSA-N Monacolin X Natural products C12C(OC(=O)C(C)CC)CC(C)C=C2C=CC(C)C1CCC1CC(O)CC(=O)O1 PCZOHLXUXFIOCF-UHFFFAOYSA-N 0.000 description 3
- 108010066427 N-valyltryptophan Proteins 0.000 description 3
- 206010028980 Neoplasm Diseases 0.000 description 3
- CSYVXYQDIVCQNU-QWRGUYRKSA-N Phe-Asp-Gly Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CC(O)=O)C(=O)NCC(O)=O CSYVXYQDIVCQNU-QWRGUYRKSA-N 0.000 description 3
- IILUKIJNFMUBNF-IHRRRGAJSA-N Phe-Gln-Gln Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCC(N)=O)C(O)=O IILUKIJNFMUBNF-IHRRRGAJSA-N 0.000 description 3
- JXQVYPWVGUOIDV-MXAVVETBSA-N Phe-Ser-Ile Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CO)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O JXQVYPWVGUOIDV-MXAVVETBSA-N 0.000 description 3
- UNBFGVQVQGXXCK-KKUMJFAQSA-N Phe-Ser-Leu Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(O)=O UNBFGVQVQGXXCK-KKUMJFAQSA-N 0.000 description 3
- 239000004698 Polyethylene Substances 0.000 description 3
- VCYJKOLZYPYGJV-AVGNSLFASA-N Pro-Arg-Leu Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(C)C)C(O)=O VCYJKOLZYPYGJV-AVGNSLFASA-N 0.000 description 3
- MGDFPGCFVJFITQ-CIUDSAMLSA-N Pro-Glu-Asp Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(O)=O)C(O)=O MGDFPGCFVJFITQ-CIUDSAMLSA-N 0.000 description 3
- LNICFEXCAHIJOR-DCAQKATOSA-N Pro-Ser-Leu Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(O)=O LNICFEXCAHIJOR-DCAQKATOSA-N 0.000 description 3
- DNIAPMSPPWPWGF-UHFFFAOYSA-N Propylene glycol Chemical compound CC(O)CO DNIAPMSPPWPWGF-UHFFFAOYSA-N 0.000 description 3
- RYMZZMVNJRMUDD-UHFFFAOYSA-N SJ000286063 Natural products C12C(OC(=O)C(C)(C)CC)CC(C)C=C2C=CC(C)C1CCC1CC(O)CC(=O)O1 RYMZZMVNJRMUDD-UHFFFAOYSA-N 0.000 description 3
- WDXYVIIVDIDOSX-DCAQKATOSA-N Ser-Arg-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)[C@@H](N)CO)CCCN=C(N)N WDXYVIIVDIDOSX-DCAQKATOSA-N 0.000 description 3
- HBOABDXGTMMDSE-GUBZILKMSA-N Ser-Arg-Val Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](C(C)C)C(O)=O HBOABDXGTMMDSE-GUBZILKMSA-N 0.000 description 3
- VGNYHOBZJKWRGI-CIUDSAMLSA-N Ser-Asn-Lys Chemical compound NCCCC[C@@H](C(O)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@@H](N)CO VGNYHOBZJKWRGI-CIUDSAMLSA-N 0.000 description 3
- ULVMNZOKDBHKKI-ACZMJKKPSA-N Ser-Gln-Asp Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(O)=O)C(O)=O ULVMNZOKDBHKKI-ACZMJKKPSA-N 0.000 description 3
- KDGARKCAKHBEDB-NKWVEPMBSA-N Ser-Gly-Pro Chemical compound C1C[C@@H](N(C1)C(=O)CNC(=O)[C@H](CO)N)C(=O)O KDGARKCAKHBEDB-NKWVEPMBSA-N 0.000 description 3
- SFTZWNJFZYOLBD-ZDLURKLDSA-N Ser-Gly-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)CNC(=O)[C@@H](N)CO SFTZWNJFZYOLBD-ZDLURKLDSA-N 0.000 description 3
- CAOYHZOWXFFAIR-CIUDSAMLSA-N Ser-His-Ser Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CO)C(O)=O CAOYHZOWXFFAIR-CIUDSAMLSA-N 0.000 description 3
- ZKBKUWQVDWWSRI-BZSNNMDCSA-N Ser-Phe-Tyr Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O ZKBKUWQVDWWSRI-BZSNNMDCSA-N 0.000 description 3
- ADJDNJCSPNFFPI-FXQIFTODSA-N Ser-Pro-Ala Chemical compound OC(=O)[C@H](C)NC(=O)[C@@H]1CCCN1C(=O)[C@@H](N)CO ADJDNJCSPNFFPI-FXQIFTODSA-N 0.000 description 3
- RHAPJNVNWDBFQI-BQBZGAKWSA-N Ser-Pro-Gly Chemical compound OC[C@H](N)C(=O)N1CCC[C@H]1C(=O)NCC(O)=O RHAPJNVNWDBFQI-BQBZGAKWSA-N 0.000 description 3
- CUXJENOFJXOSOZ-BIIVOSGPSA-N Ser-Ser-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CO)NC(=O)[C@H](CO)N)C(=O)O CUXJENOFJXOSOZ-BIIVOSGPSA-N 0.000 description 3
- DKGRNFUXVTYRAS-UBHSHLNASA-N Ser-Ser-Trp Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(O)=O DKGRNFUXVTYRAS-UBHSHLNASA-N 0.000 description 3
- RXUOAOOZIWABBW-XGEHTFHBSA-N Ser-Thr-Arg Chemical compound OC[C@H](N)C(=O)N[C@@H]([C@H](O)C)C(=O)N[C@H](C(O)=O)CCCN=C(N)N RXUOAOOZIWABBW-XGEHTFHBSA-N 0.000 description 3
- OSFZCEQJLWCIBG-BZSNNMDCSA-N Ser-Tyr-Tyr Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O OSFZCEQJLWCIBG-BZSNNMDCSA-N 0.000 description 3
- LVHHEVGYAZGXDE-KDXUFGMBSA-N Thr-Ala-Pro Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](C)C(=O)N1CCC[C@@H]1C(=O)O)N)O LVHHEVGYAZGXDE-KDXUFGMBSA-N 0.000 description 3
- WPSKTVVMQCXPRO-BWBBJGPYSA-N Thr-Ser-Ser Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CO)C(O)=O WPSKTVVMQCXPRO-BWBBJGPYSA-N 0.000 description 3
- XVHAUVJXBFGUPC-RPTUDFQQSA-N Thr-Tyr-Phe Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O XVHAUVJXBFGUPC-RPTUDFQQSA-N 0.000 description 3
- DIHPMRTXPYMDJZ-KAOXEZKKSA-N Thr-Tyr-Pro Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)N2CCC[C@@H]2C(=O)O)N)O DIHPMRTXPYMDJZ-KAOXEZKKSA-N 0.000 description 3
- CYCGARJWIQWPQM-YJRXYDGGSA-N Thr-Tyr-Ser Chemical compound C[C@@H](O)[C@H]([NH3+])C(=O)N[C@H](C(=O)N[C@@H](CO)C([O-])=O)CC1=CC=C(O)C=C1 CYCGARJWIQWPQM-YJRXYDGGSA-N 0.000 description 3
- KZTLZZQTJMCGIP-ZJDVBMNYSA-N Thr-Val-Thr Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O KZTLZZQTJMCGIP-ZJDVBMNYSA-N 0.000 description 3
- GQHAIUPYZPTADF-FDARSICLSA-N Trp-Ile-Arg Chemical compound C1=CC=C2C(C[C@H](N)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CCCN=C(N)N)C(O)=O)=CNC2=C1 GQHAIUPYZPTADF-FDARSICLSA-N 0.000 description 3
- HJWLQSFTGDQSRX-BPUTZDHNSA-N Trp-Met-Ser Chemical compound [H]N[C@@H](CC1=CNC2=C1C=CC=C2)C(=O)N[C@@H](CCSC)C(=O)N[C@@H](CO)C(O)=O HJWLQSFTGDQSRX-BPUTZDHNSA-N 0.000 description 3
- UJGDFQRPYGJBEH-AAEUAGOBSA-N Trp-Ser-Gly Chemical compound C1=CC=C2C(=C1)C(=CN2)C[C@@H](C(=O)N[C@@H](CO)C(=O)NCC(=O)O)N UJGDFQRPYGJBEH-AAEUAGOBSA-N 0.000 description 3
- PKZIWSHDJYIPRH-JBACZVJFSA-N Trp-Tyr-Gln Chemical compound [H]N[C@@H](CC1=CNC2=C1C=CC=C2)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CCC(N)=O)C(O)=O PKZIWSHDJYIPRH-JBACZVJFSA-N 0.000 description 3
- SMLCYZYQFRTLCO-UWJYBYFXSA-N Tyr-Cys-Ala Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CS)C(=O)N[C@@H](C)C(O)=O SMLCYZYQFRTLCO-UWJYBYFXSA-N 0.000 description 3
- YLRLHDFMMWDYTK-KKUMJFAQSA-N Tyr-Cys-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CS)NC(=O)[C@@H](N)CC1=CC=C(O)C=C1 YLRLHDFMMWDYTK-KKUMJFAQSA-N 0.000 description 3
- QHEGAOPHISYNDF-XDTLVQLUSA-N Tyr-Gln-Ala Chemical compound C[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CC1=CC=C(C=C1)O)N QHEGAOPHISYNDF-XDTLVQLUSA-N 0.000 description 3
- NQJDICVXXIMMMB-XDTLVQLUSA-N Tyr-Glu-Ala Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](C)C(O)=O NQJDICVXXIMMMB-XDTLVQLUSA-N 0.000 description 3
- CNLKDWSAORJEMW-KWQFWETISA-N Tyr-Gly-Ala Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)NCC(=O)N[C@@H](C)C(O)=O CNLKDWSAORJEMW-KWQFWETISA-N 0.000 description 3
- OVBMCNDKCWAXMZ-NAKRPEOUSA-N Val-Ile-Ser Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CO)C(=O)O)NC(=O)[C@H](C(C)C)N OVBMCNDKCWAXMZ-NAKRPEOUSA-N 0.000 description 3
- MYLNLEIZWHVENT-VKOGCVSHSA-N Val-Ile-Trp Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC1=CNC2=CC=CC=C21)C(=O)O)NC(=O)[C@H](C(C)C)N MYLNLEIZWHVENT-VKOGCVSHSA-N 0.000 description 3
- GBIUHAYJGWVNLN-AEJSXWLSSA-N Val-Ser-Pro Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CO)C(=O)N1CCC[C@@H]1C(=O)O)N GBIUHAYJGWVNLN-AEJSXWLSSA-N 0.000 description 3
- GBIUHAYJGWVNLN-UHFFFAOYSA-N Val-Ser-Pro Natural products CC(C)C(N)C(=O)NC(CO)C(=O)N1CCCC1C(O)=O GBIUHAYJGWVNLN-UHFFFAOYSA-N 0.000 description 3
- 230000033115 angiogenesis Effects 0.000 description 3
- 239000004037 angiogenesis inhibitor Substances 0.000 description 3
- 108010077245 asparaginyl-proline Proteins 0.000 description 3
- 108010040443 aspartyl-aspartic acid Proteins 0.000 description 3
- 230000000903 blocking effect Effects 0.000 description 3
- 230000037396 body weight Effects 0.000 description 3
- 230000007812 deficiency Effects 0.000 description 3
- 230000006870 function Effects 0.000 description 3
- HNDVDQJCIGZPNO-UHFFFAOYSA-N histidine Natural products OC(=O)C(N)CC1=CN=CN1 HNDVDQJCIGZPNO-UHFFFAOYSA-N 0.000 description 3
- 108010018006 histidylserine Proteins 0.000 description 3
- 238000000338 in vitro Methods 0.000 description 3
- 238000003780 insertion Methods 0.000 description 3
- 230000037431 insertion Effects 0.000 description 3
- 230000003993 interaction Effects 0.000 description 3
- 238000012933 kinetic analysis Methods 0.000 description 3
- 108010090333 leucyl-lysyl-proline Proteins 0.000 description 3
- 239000002502 liposome Substances 0.000 description 3
- 229960004844 lovastatin Drugs 0.000 description 3
- PCZOHLXUXFIOCF-BXMDZJJMSA-N lovastatin Chemical compound C([C@H]1[C@@H](C)C=CC2=C[C@H](C)C[C@@H]([C@H]12)OC(=O)[C@@H](C)CC)C[C@@H]1C[C@@H](O)CC(=O)O1 PCZOHLXUXFIOCF-BXMDZJJMSA-N 0.000 description 3
- QLJODMDSTUBWDW-UHFFFAOYSA-N lovastatin hydroxy acid Natural products C1=CC(C)C(CCC(O)CC(O)CC(O)=O)C2C(OC(=O)C(C)CC)CC(C)C=C21 QLJODMDSTUBWDW-UHFFFAOYSA-N 0.000 description 3
- 239000000463 material Substances 0.000 description 3
- 239000011159 matrix material Substances 0.000 description 3
- 230000007246 mechanism Effects 0.000 description 3
- 230000003285 pharmacodynamic effect Effects 0.000 description 3
- ORMNNUPLFAPCFD-DVLYDCSHSA-M phenethicillin potassium Chemical compound [K+].N([C@@H]1C(N2[C@H](C(C)(C)S[C@@H]21)C([O-])=O)=O)C(=O)C(C)OC1=CC=CC=C1 ORMNNUPLFAPCFD-DVLYDCSHSA-M 0.000 description 3
- 125000002924 primary amino group Chemical group [H]N([H])* 0.000 description 3
- 230000002285 radioactive effect Effects 0.000 description 3
- 238000003127 radioimmunoassay Methods 0.000 description 3
- 102220117530 rs112626848 Human genes 0.000 description 3
- 102220238658 rs1468529365 Human genes 0.000 description 3
- 102220268018 rs201210997 Human genes 0.000 description 3
- 239000000523 sample Substances 0.000 description 3
- 238000002864 sequence alignment Methods 0.000 description 3
- 229960002855 simvastatin Drugs 0.000 description 3
- RYMZZMVNJRMUDD-HGQWONQESA-N simvastatin Chemical compound C([C@H]1[C@@H](C)C=CC2=C[C@H](C)C[C@@H]([C@H]12)OC(=O)C(C)(C)CC)C[C@@H]1C[C@@H](O)CC(=O)O1 RYMZZMVNJRMUDD-HGQWONQESA-N 0.000 description 3
- 108010072986 threonyl-seryl-lysine Proteins 0.000 description 3
- 108010017949 tyrosyl-glycyl-glycine Proteins 0.000 description 3
- ONDPHDOFVYQSGI-UHFFFAOYSA-N zinc nitrate Chemical compound [Zn+2].[O-][N+]([O-])=O.[O-][N+]([O-])=O ONDPHDOFVYQSGI-UHFFFAOYSA-N 0.000 description 3
- FJLGEFLZQAZZCD-MCBHFWOFSA-N (3R,5S)-fluvastatin Chemical compound C12=CC=CC=C2N(C(C)C)C(\C=C\[C@@H](O)C[C@@H](O)CC(O)=O)=C1C1=CC=C(F)C=C1 FJLGEFLZQAZZCD-MCBHFWOFSA-N 0.000 description 2
- CABVTRNMFUVUDM-VRHQGPGLSA-N (3S)-3-hydroxy-3-methylglutaryl-CoA Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCSC(=O)C[C@@](O)(CC(O)=O)C)O[C@H]1N1C2=NC=NC(N)=C2N=C1 CABVTRNMFUVUDM-VRHQGPGLSA-N 0.000 description 2
- SRSGVKWWVXWSJT-ATVHPVEESA-N 5-[(z)-(5-fluoro-2-oxo-1h-indol-3-ylidene)methyl]-2,4-dimethyl-n-(2-pyrrolidin-1-ylethyl)-1h-pyrrole-3-carboxamide Chemical compound CC=1NC(\C=C/2C3=CC(F)=CC=C3NC\2=O)=C(C)C=1C(=O)NCCN1CCCC1 SRSGVKWWVXWSJT-ATVHPVEESA-N 0.000 description 2
- XQJAFSDFQZPYCU-UWJYBYFXSA-N Ala-Asn-Tyr Chemical compound C[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)O)N XQJAFSDFQZPYCU-UWJYBYFXSA-N 0.000 description 2
- DVJSJDDYCYSMFR-ZKWXMUAHSA-N Ala-Ile-Gly Chemical compound [H]N[C@@H](C)C(=O)N[C@@H]([C@@H](C)CC)C(=O)NCC(O)=O DVJSJDDYCYSMFR-ZKWXMUAHSA-N 0.000 description 2
- WUHJHHGYVVJMQE-BJDJZHNGSA-N Ala-Leu-Ile Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O WUHJHHGYVVJMQE-BJDJZHNGSA-N 0.000 description 2
- RAAWHFXHAACDFT-FXQIFTODSA-N Ala-Met-Asn Chemical compound CSCC[C@H](NC(=O)[C@H](C)N)C(=O)N[C@@H](CC(N)=O)C(O)=O RAAWHFXHAACDFT-FXQIFTODSA-N 0.000 description 2
- DYXOFPBJBAHWFY-JBDRJPRFSA-N Ala-Ser-Ile Chemical compound CC[C@H](C)[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@H](C)N DYXOFPBJBAHWFY-JBDRJPRFSA-N 0.000 description 2
- SYIFFFHSXBNPMC-UWJYBYFXSA-N Ala-Ser-Tyr Chemical compound C[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)O)N SYIFFFHSXBNPMC-UWJYBYFXSA-N 0.000 description 2
- OMSKGWFGWCQFBD-KZVJFYERSA-N Ala-Val-Thr Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O OMSKGWFGWCQFBD-KZVJFYERSA-N 0.000 description 2
- XKJMBINCVNINCA-UHFFFAOYSA-N Alfalone Chemical compound CON(C)C(=O)NC1=CC=C(Cl)C(Cl)=C1 XKJMBINCVNINCA-UHFFFAOYSA-N 0.000 description 2
- 102100034567 Angiopoietin-related protein 5 Human genes 0.000 description 2
- 102100034599 Angiopoietin-related protein 6 Human genes 0.000 description 2
- NTAZNGWBXRVEDJ-FXQIFTODSA-N Arg-Asp-Asp Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC(O)=O)C(O)=O NTAZNGWBXRVEDJ-FXQIFTODSA-N 0.000 description 2
- KRQSPVKUISQQFS-FJXKBIBVSA-N Arg-Gly-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)CNC(=O)[C@@H](N)CCCN=C(N)N KRQSPVKUISQQFS-FJXKBIBVSA-N 0.000 description 2
- OVQJAKFLFTZDNC-GUBZILKMSA-N Arg-Pro-Asp Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CC(O)=O)C(O)=O OVQJAKFLFTZDNC-GUBZILKMSA-N 0.000 description 2
- 239000004475 Arginine Substances 0.000 description 2
- ZZXMOQIUIJJOKZ-ZLUOBGJFSA-N Asn-Asn-Ala Chemical compound OC(=O)[C@H](C)NC(=O)[C@H](CC(N)=O)NC(=O)[C@@H](N)CC(N)=O ZZXMOQIUIJJOKZ-ZLUOBGJFSA-N 0.000 description 2
- HFPXZWPUVFVNLL-GUBZILKMSA-N Asn-Leu-Gln Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(N)=O)C(O)=O HFPXZWPUVFVNLL-GUBZILKMSA-N 0.000 description 2
- PBFXCUOEGVJTMV-QXEWZRGKSA-N Asn-Met-Val Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CCSC)C(=O)N[C@@H](C(C)C)C(O)=O PBFXCUOEGVJTMV-QXEWZRGKSA-N 0.000 description 2
- HPBNLFLSSQDFQW-WHFBIAKZSA-N Asn-Ser-Gly Chemical compound NC(=O)C[C@H](N)C(=O)N[C@@H](CO)C(=O)NCC(O)=O HPBNLFLSSQDFQW-WHFBIAKZSA-N 0.000 description 2
- MYTHOBCLNIOFBL-SRVKXCTJSA-N Asn-Ser-Tyr Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O MYTHOBCLNIOFBL-SRVKXCTJSA-N 0.000 description 2
- SNDBKTFJWVEVPO-WHFBIAKZSA-N Asp-Gly-Ser Chemical compound [H]N[C@@H](CC(O)=O)C(=O)NCC(=O)N[C@@H](CO)C(O)=O SNDBKTFJWVEVPO-WHFBIAKZSA-N 0.000 description 2
- QNMKWNONJGKJJC-NHCYSSNCSA-N Asp-Leu-Val Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](C(C)C)C(O)=O QNMKWNONJGKJJC-NHCYSSNCSA-N 0.000 description 2
- XWSIYTYNLKCLJB-CIUDSAMLSA-N Asp-Lys-Asn Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(N)=O)C(O)=O XWSIYTYNLKCLJB-CIUDSAMLSA-N 0.000 description 2
- VMVUDJUXJKDGNR-FXQIFTODSA-N Asp-Met-Asn Chemical compound CSCC[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)O)NC(=O)[C@H](CC(=O)O)N VMVUDJUXJKDGNR-FXQIFTODSA-N 0.000 description 2
- JDDYEZGPYBBPBN-JRQIVUDYSA-N Asp-Thr-Tyr Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O JDDYEZGPYBBPBN-JRQIVUDYSA-N 0.000 description 2
- PLNJUJGNLDSFOP-UWJYBYFXSA-N Asp-Tyr-Ala Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](C)C(O)=O PLNJUJGNLDSFOP-UWJYBYFXSA-N 0.000 description 2
- XUKUURHRXDUEBC-KAYWLYCHSA-N Atorvastatin Chemical compound C=1C=CC=CC=1C1=C(C=2C=CC(F)=CC=2)N(CC[C@@H](O)C[C@@H](O)CC(O)=O)C(C(C)C)=C1C(=O)NC1=CC=CC=C1 XUKUURHRXDUEBC-KAYWLYCHSA-N 0.000 description 2
- XUKUURHRXDUEBC-UHFFFAOYSA-N Atorvastatin Natural products C=1C=CC=CC=1C1=C(C=2C=CC(F)=CC=2)N(CCC(O)CC(O)CC(O)=O)C(C(C)C)=C1C(=O)NC1=CC=CC=C1 XUKUURHRXDUEBC-UHFFFAOYSA-N 0.000 description 2
- MLDQJTXFUGDVEO-UHFFFAOYSA-N BAY-43-9006 Chemical compound C1=NC(C(=O)NC)=CC(OC=2C=CC(NC(=O)NC=3C=C(C(Cl)=CC=3)C(F)(F)F)=CC=2)=C1 MLDQJTXFUGDVEO-UHFFFAOYSA-N 0.000 description 2
- AIRYAONNMGRCGJ-FHFVDXKLSA-N CC[C@H](C)[C@H](NC(=O)CN)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@H]1CSSC[C@@H]2NC(=O)[C@@H](NC(=O)[C@H](CO)NC(=O)[C@@H](NC(=O)[C@H](CSSC[C@H](NC(=O)[C@H](CC(C)C)NC(=O)[C@H](Cc3c[nH]cn3)NC(=O)[C@H](CCC(N)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@@H](NC(=O)[C@@H](N)Cc3ccccc3)C(C)C)C(=O)NCC(=O)N[C@@H](CO)C(=O)N[C@@H](Cc3c[nH]cn3)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](C)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](Cc3ccc(O)cc3)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CSSC[C@H](NC(=O)[C@H](Cc3ccc(O)cc3)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CCC(N)=O)NC(=O)[C@H](Cc3ccc(O)cc3)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CO)NC2=O)C(=O)N[C@@H](CC(N)=O)C(O)=O)C(=O)NCC(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)NCC(=O)N[C@@H](Cc2ccccc2)C(=O)N[C@@H](Cc2ccccc2)C(=O)N[C@@H](Cc2ccc(O)cc2)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCCCN)C(=O)N2CCC[C@H]2C(=O)N[C@@H]([C@@H](C)O)C(O)=O)NC1=O)[C@@H](C)O)[C@@H](C)CC Chemical compound CC[C@H](C)[C@H](NC(=O)CN)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@H]1CSSC[C@@H]2NC(=O)[C@@H](NC(=O)[C@H](CO)NC(=O)[C@@H](NC(=O)[C@H](CSSC[C@H](NC(=O)[C@H](CC(C)C)NC(=O)[C@H](Cc3c[nH]cn3)NC(=O)[C@H](CCC(N)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@@H](NC(=O)[C@@H](N)Cc3ccccc3)C(C)C)C(=O)NCC(=O)N[C@@H](CO)C(=O)N[C@@H](Cc3c[nH]cn3)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](C)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](Cc3ccc(O)cc3)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CSSC[C@H](NC(=O)[C@H](Cc3ccc(O)cc3)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CCC(N)=O)NC(=O)[C@H](Cc3ccc(O)cc3)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CO)NC2=O)C(=O)N[C@@H](CC(N)=O)C(O)=O)C(=O)NCC(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)NCC(=O)N[C@@H](Cc2ccccc2)C(=O)N[C@@H](Cc2ccccc2)C(=O)N[C@@H](Cc2ccc(O)cc2)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCCCN)C(=O)N2CCC[C@H]2C(=O)N[C@@H]([C@@H](C)O)C(O)=O)NC1=O)[C@@H](C)O)[C@@H](C)CC AIRYAONNMGRCGJ-FHFVDXKLSA-N 0.000 description 2
- 101710112752 Cytotoxin Proteins 0.000 description 2
- 108700024394 Exon Proteins 0.000 description 2
- WUAYFMZULZDSLB-ACZMJKKPSA-N Gln-Ala-Asn Chemical compound NC(=O)C[C@@H](C(O)=O)NC(=O)[C@H](C)NC(=O)[C@@H](N)CCC(N)=O WUAYFMZULZDSLB-ACZMJKKPSA-N 0.000 description 2
- KVXVVDFOZNYYKZ-DCAQKATOSA-N Gln-Gln-Leu Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(C)C)C(O)=O KVXVVDFOZNYYKZ-DCAQKATOSA-N 0.000 description 2
- FGYPOQPQTUNESW-IUCAKERBSA-N Gln-Gly-Leu Chemical compound CC(C)C[C@@H](C(=O)O)NC(=O)CNC(=O)[C@H](CCC(=O)N)N FGYPOQPQTUNESW-IUCAKERBSA-N 0.000 description 2
- ZBKUIQNCRIYVGH-SDDRHHMPSA-N Gln-Leu-Pro Chemical compound CC(C)C[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CCC(=O)N)N ZBKUIQNCRIYVGH-SDDRHHMPSA-N 0.000 description 2
- KPNWAJMEMRCLAL-GUBZILKMSA-N Gln-Ser-Lys Chemical compound C(CCN)C[C@@H](C(=O)O)NC(=O)[C@H](CO)NC(=O)[C@H](CCC(=O)N)N KPNWAJMEMRCLAL-GUBZILKMSA-N 0.000 description 2
- MKRDNSWGJWTBKZ-GVXVVHGQSA-N Gln-Val-Lys Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CCC(=O)N)N MKRDNSWGJWTBKZ-GVXVVHGQSA-N 0.000 description 2
- HNAUFGBKJLTWQE-IFFSRLJSSA-N Gln-Val-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](C(C)C)NC(=O)[C@H](CCC(=O)N)N)O HNAUFGBKJLTWQE-IFFSRLJSSA-N 0.000 description 2
- UTKUTMJSWKKHEM-WDSKDSINSA-N Glu-Ala-Gly Chemical compound OC(=O)CNC(=O)[C@H](C)NC(=O)[C@@H](N)CCC(O)=O UTKUTMJSWKKHEM-WDSKDSINSA-N 0.000 description 2
- DSPQRJXOIXHOHK-WDSKDSINSA-N Glu-Asp-Gly Chemical compound OC(=O)CC[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)NCC(O)=O DSPQRJXOIXHOHK-WDSKDSINSA-N 0.000 description 2
- CYHBMLHCQXXCCT-AVGNSLFASA-N Glu-Asp-Tyr Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O CYHBMLHCQXXCCT-AVGNSLFASA-N 0.000 description 2
- QCMVGXDELYMZET-GLLZPBPUSA-N Glu-Thr-Glu Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCC(O)=O)C(O)=O QCMVGXDELYMZET-GLLZPBPUSA-N 0.000 description 2
- HHSOPSCKAZKQHQ-PEXQALLHSA-N Gly-His-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H](CC1=CN=CN1)NC(=O)CN HHSOPSCKAZKQHQ-PEXQALLHSA-N 0.000 description 2
- DENRBIYENOKSEX-PEXQALLHSA-N Gly-Ile-His Chemical compound NCC(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@H](C(O)=O)CC1=CN=CN1 DENRBIYENOKSEX-PEXQALLHSA-N 0.000 description 2
- PEDCQBHIVMGVHV-UHFFFAOYSA-N Glycerol Natural products OCC(O)CO PEDCQBHIVMGVHV-UHFFFAOYSA-N 0.000 description 2
- 108010023302 HDL Cholesterol Proteins 0.000 description 2
- IDQKGZWUPVOGPZ-GUBZILKMSA-N His-Cys-Gln Chemical compound C1=C(NC=N1)C[C@@H](C(=O)N[C@@H](CS)C(=O)N[C@@H](CCC(=O)N)C(=O)O)N IDQKGZWUPVOGPZ-GUBZILKMSA-N 0.000 description 2
- LNDVNHOSZQPJGI-AVGNSLFASA-N His-Pro-Pro Chemical compound C([C@H](N)C(=O)N1[C@@H](CCC1)C(=O)N1[C@@H](CCC1)C(O)=O)C1=CN=CN1 LNDVNHOSZQPJGI-AVGNSLFASA-N 0.000 description 2
- YERBCFWVWITTEJ-NAZCDGGXSA-N His-Trp-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CC1=CNC2=CC=CC=C21)NC(=O)[C@H](CC3=CN=CN3)N)O YERBCFWVWITTEJ-NAZCDGGXSA-N 0.000 description 2
- 101000693085 Homo sapiens Angiopoietin-related protein 3 Proteins 0.000 description 2
- 101000924346 Homo sapiens Angiopoietin-related protein 5 Proteins 0.000 description 2
- 101000924549 Homo sapiens Angiopoietin-related protein 6 Proteins 0.000 description 2
- 101001098868 Homo sapiens Proprotein convertase subtilisin/kexin type 9 Proteins 0.000 description 2
- JXMSHKFPDIUYGS-SIUGBPQLSA-N Ile-Glu-Tyr Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CCC(=O)O)C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)O)N JXMSHKFPDIUYGS-SIUGBPQLSA-N 0.000 description 2
- LBRCLQMZAHRTLV-ZKWXMUAHSA-N Ile-Gly-Ser Chemical compound CC[C@H](C)[C@H](N)C(=O)NCC(=O)N[C@@H](CO)C(O)=O LBRCLQMZAHRTLV-ZKWXMUAHSA-N 0.000 description 2
- UAQSZXGJGLHMNV-XEGUGMAKSA-N Ile-Gly-Tyr Chemical compound CC[C@H](C)[C@@H](C(=O)NCC(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)O)N UAQSZXGJGLHMNV-XEGUGMAKSA-N 0.000 description 2
- ZNOBVZFCHNHKHA-KBIXCLLPSA-N Ile-Ser-Glu Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CCC(=O)O)C(=O)O)N ZNOBVZFCHNHKHA-KBIXCLLPSA-N 0.000 description 2
- JDCQDJVYUXNCGF-SPOWBLRKSA-N Ile-Ser-Trp Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CC1=CNC2=CC=CC=C21)C(=O)O)N JDCQDJVYUXNCGF-SPOWBLRKSA-N 0.000 description 2
- QSXSHZIRKTUXNG-STECZYCISA-N Ile-Val-Tyr Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 QSXSHZIRKTUXNG-STECZYCISA-N 0.000 description 2
- 101710172072 Kexin Proteins 0.000 description 2
- IBMVEYRWAWIOTN-UHFFFAOYSA-N L-Leucyl-L-Arginyl-L-Proline Natural products CC(C)CC(N)C(=O)NC(CCCN=C(N)N)C(=O)N1CCCC1C(O)=O IBMVEYRWAWIOTN-UHFFFAOYSA-N 0.000 description 2
- WHUUTDBJXJRKMK-VKHMYHEASA-N L-glutamic acid Chemical compound OC(=O)[C@@H](N)CCC(O)=O WHUUTDBJXJRKMK-VKHMYHEASA-N 0.000 description 2
- AGPKZVBTJJNPAG-WHFBIAKZSA-N L-isoleucine Chemical compound CC[C@H](C)[C@H](N)C(O)=O AGPKZVBTJJNPAG-WHFBIAKZSA-N 0.000 description 2
- LHSGPCFBGJHPCY-UHFFFAOYSA-N L-leucine-L-tyrosine Natural products CC(C)CC(N)C(=O)NC(C(O)=O)CC1=CC=C(O)C=C1 LHSGPCFBGJHPCY-UHFFFAOYSA-N 0.000 description 2
- 239000002147 L01XE04 - Sunitinib Substances 0.000 description 2
- 239000005511 L01XE05 - Sorafenib Substances 0.000 description 2
- 239000003798 L01XE11 - Pazopanib Substances 0.000 description 2
- 102000000853 LDL receptors Human genes 0.000 description 2
- 108010001831 LDL receptors Proteins 0.000 description 2
- KAFOIVJDVSZUMD-DCAQKATOSA-N Leu-Gln-Gln Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCC(N)=O)C(O)=O KAFOIVJDVSZUMD-DCAQKATOSA-N 0.000 description 2
- KAFOIVJDVSZUMD-UHFFFAOYSA-N Leu-Gln-Gln Natural products CC(C)CC(N)C(=O)NC(CCC(N)=O)C(=O)NC(CCC(N)=O)C(O)=O KAFOIVJDVSZUMD-UHFFFAOYSA-N 0.000 description 2
- ZTLGVASZOIKNIX-DCAQKATOSA-N Leu-Gln-Glu Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)N[C@@H](CCC(=O)O)C(=O)O)N ZTLGVASZOIKNIX-DCAQKATOSA-N 0.000 description 2
- JGKHAFUAPZCCDU-BZSNNMDCSA-N Leu-Tyr-Leu Chemical compound CC(C)C[C@H]([NH3+])C(=O)N[C@H](C(=O)N[C@@H](CC(C)C)C([O-])=O)CC1=CC=C(O)C=C1 JGKHAFUAPZCCDU-BZSNNMDCSA-N 0.000 description 2
- NTXYXFDMIHXTHE-WDSOQIARSA-N Leu-Val-Trp Chemical compound C1=CC=C2C(C[C@H](NC(=O)[C@H](C(C)C)NC(=O)[C@@H](N)CC(C)C)C(O)=O)=CNC2=C1 NTXYXFDMIHXTHE-WDSOQIARSA-N 0.000 description 2
- 102000004882 Lipase Human genes 0.000 description 2
- 239000004367 Lipase Substances 0.000 description 2
- 108090001060 Lipase Proteins 0.000 description 2
- FLCMXEFCTLXBTL-DCAQKATOSA-N Lys-Asp-Arg Chemical compound C(CCN)C[C@@H](C(=O)N[C@@H](CC(=O)O)C(=O)N[C@@H](CCCN=C(N)N)C(=O)O)N FLCMXEFCTLXBTL-DCAQKATOSA-N 0.000 description 2
- LUAJJLPHUXPQLH-KKUMJFAQSA-N Lys-Phe-Ser Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CO)C(=O)O)NC(=O)[C@H](CCCCN)N LUAJJLPHUXPQLH-KKUMJFAQSA-N 0.000 description 2
- SBQDRNOLGSYHQA-YUMQZZPRSA-N Lys-Ser-Gly Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CO)C(=O)NCC(O)=O SBQDRNOLGSYHQA-YUMQZZPRSA-N 0.000 description 2
- 241000124008 Mammalia Species 0.000 description 2
- 208000001145 Metabolic Syndrome Diseases 0.000 description 2
- MSSXKZBDKZAHCX-UNQGMJICSA-N Phe-Thr-Val Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C(C)C)C(O)=O MSSXKZBDKZAHCX-UNQGMJICSA-N 0.000 description 2
- 206010035226 Plasma cell myeloma Diseases 0.000 description 2
- TUZYXOIXSAXUGO-UHFFFAOYSA-N Pravastatin Natural products C1=CC(C)C(CCC(O)CC(O)CC(O)=O)C2C(OC(=O)C(C)CC)CC(O)C=C21 TUZYXOIXSAXUGO-UHFFFAOYSA-N 0.000 description 2
- VTFXTWDFPTWNJY-RHYQMDGZSA-N Pro-Leu-Thr Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O VTFXTWDFPTWNJY-RHYQMDGZSA-N 0.000 description 2
- IIRBTQHFVNGPMQ-AVGNSLFASA-N Pro-Val-Lys Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@@H]1CCCN1 IIRBTQHFVNGPMQ-AVGNSLFASA-N 0.000 description 2
- WDVSHHCDHLJJJR-UHFFFAOYSA-N Proflavine Chemical compound C1=CC(N)=CC2=NC3=CC(N)=CC=C3C=C21 WDVSHHCDHLJJJR-UHFFFAOYSA-N 0.000 description 2
- 102100038955 Proprotein convertase subtilisin/kexin type 9 Human genes 0.000 description 2
- 239000004365 Protease Substances 0.000 description 2
- HRNQLKCLPVKZNE-CIUDSAMLSA-N Ser-Ala-Leu Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](C)C(=O)N[C@@H](CC(C)C)C(O)=O HRNQLKCLPVKZNE-CIUDSAMLSA-N 0.000 description 2
- UBRMZSHOOIVJPW-SRVKXCTJSA-N Ser-Leu-Lys Chemical compound OC[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(O)=O UBRMZSHOOIVJPW-SRVKXCTJSA-N 0.000 description 2
- RWDVVSKYZBNDCO-MELADBBJSA-N Ser-Phe-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CC2=CC=CC=C2)NC(=O)[C@H](CO)N)C(=O)O RWDVVSKYZBNDCO-MELADBBJSA-N 0.000 description 2
- OLKICIBQRVSQMA-SRVKXCTJSA-N Ser-Ser-Tyr Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O OLKICIBQRVSQMA-SRVKXCTJSA-N 0.000 description 2
- MFQMZDPAZRZAPV-NAKRPEOUSA-N Ser-Val-Ile Chemical compound CC[C@H](C)[C@@H](C(=O)O)NC(=O)[C@H](C(C)C)NC(=O)[C@H](CO)N MFQMZDPAZRZAPV-NAKRPEOUSA-N 0.000 description 2
- LGIMRDKGABDMBN-DCAQKATOSA-N Ser-Val-Lys Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CO)N LGIMRDKGABDMBN-DCAQKATOSA-N 0.000 description 2
- 108090000787 Subtilisin Proteins 0.000 description 2
- NJEMRSFGDNECGF-GCJQMDKQSA-N Thr-Ala-Asp Chemical compound C[C@@H](O)[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@H](C(O)=O)CC(O)=O NJEMRSFGDNECGF-GCJQMDKQSA-N 0.000 description 2
- TYVAWPFQYFPSBR-BFHQHQDPSA-N Thr-Ala-Gly Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C)C(=O)NCC(O)=O TYVAWPFQYFPSBR-BFHQHQDPSA-N 0.000 description 2
- BSNZTJXVDOINSR-JXUBOQSCSA-N Thr-Ala-Leu Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C)C(=O)N[C@@H](CC(C)C)C(O)=O BSNZTJXVDOINSR-JXUBOQSCSA-N 0.000 description 2
- JEDIEMIJYSRUBB-FOHZUACHSA-N Thr-Asp-Gly Chemical compound C[C@@H](O)[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)NCC(O)=O JEDIEMIJYSRUBB-FOHZUACHSA-N 0.000 description 2
- LGNBRHZANHMZHK-NUMRIWBASA-N Thr-Glu-Asp Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CCC(=O)O)C(=O)N[C@@H](CC(=O)O)C(=O)O)N)O LGNBRHZANHMZHK-NUMRIWBASA-N 0.000 description 2
- VRUFCJZQDACGLH-UVOCVTCTSA-N Thr-Leu-Thr Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O VRUFCJZQDACGLH-UVOCVTCTSA-N 0.000 description 2
- DXPURPNJDFCKKO-RHYQMDGZSA-N Thr-Lys-Val Chemical compound CC(C)[C@H](NC(=O)[C@H](CCCCN)NC(=O)[C@@H](N)[C@@H](C)O)C(O)=O DXPURPNJDFCKKO-RHYQMDGZSA-N 0.000 description 2
- MXNAOGFNFNKUPD-JHYOHUSXSA-N Thr-Phe-Thr Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H]([C@@H](C)O)C(O)=O MXNAOGFNFNKUPD-JHYOHUSXSA-N 0.000 description 2
- BPGDJSUFQKWUBK-KJEVXHAQSA-N Thr-Val-Tyr Chemical compound C[C@@H](O)[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 BPGDJSUFQKWUBK-KJEVXHAQSA-N 0.000 description 2
- 239000003819 Toceranib Substances 0.000 description 2
- FEZASNVQLJQBHW-CABZTGNLSA-N Trp-Gly-Ala Chemical compound C1=CC=C2C(C[C@H](N)C(=O)NCC(=O)N[C@@H](C)C(O)=O)=CNC2=C1 FEZASNVQLJQBHW-CABZTGNLSA-N 0.000 description 2
- VFJIWSJKZJTQII-SRVKXCTJSA-N Tyr-Asp-Ser Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CO)C(O)=O VFJIWSJKZJTQII-SRVKXCTJSA-N 0.000 description 2
- UMXSDHPSMROQRB-YJRXYDGGSA-N Tyr-Cys-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CS)NC(=O)[C@H](CC1=CC=C(C=C1)O)N)O UMXSDHPSMROQRB-YJRXYDGGSA-N 0.000 description 2
- IYHNBRUWVBIVJR-IHRRRGAJSA-N Tyr-Gln-Glu Chemical compound OC(=O)CC[C@@H](C(O)=O)NC(=O)[C@H](CCC(N)=O)NC(=O)[C@@H](N)CC1=CC=C(O)C=C1 IYHNBRUWVBIVJR-IHRRRGAJSA-N 0.000 description 2
- IGXLNVIYDYONFB-UFYCRDLUSA-N Tyr-Phe-Arg Chemical compound C([C@H](N)C(=O)N[C@@H](CC=1C=CC=CC=1)C(=O)N[C@@H](CCCN=C(N)N)C(O)=O)C1=CC=C(O)C=C1 IGXLNVIYDYONFB-UFYCRDLUSA-N 0.000 description 2
- RGYCVIZZTUBSSG-JYJNAYRXSA-N Tyr-Pro-Val Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N1CCC[C@H]1C(=O)N[C@@H](C(C)C)C(O)=O RGYCVIZZTUBSSG-JYJNAYRXSA-N 0.000 description 2
- MWUYSCVVPVITMW-IGNZVWTISA-N Tyr-Tyr-Ala Chemical compound C([C@@H](C(=O)N[C@@H](C)C(O)=O)NC(=O)[C@@H](N)CC=1C=CC(O)=CC=1)C1=CC=C(O)C=C1 MWUYSCVVPVITMW-IGNZVWTISA-N 0.000 description 2
- DJSYPCWZPNHQQE-FHWLQOOXSA-N Tyr-Tyr-Gln Chemical compound C([C@H](N)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(=O)N[C@@H](CCC(N)=O)C(O)=O)C1=CC=C(O)C=C1 DJSYPCWZPNHQQE-FHWLQOOXSA-N 0.000 description 2
- COSLEEOIYRPTHD-YDHLFZDLSA-N Val-Asp-Tyr Chemical compound CC(C)[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 COSLEEOIYRPTHD-YDHLFZDLSA-N 0.000 description 2
- VBTFUDNTMCHPII-UHFFFAOYSA-N Val-Trp-Tyr Natural products C=1NC2=CC=CC=C2C=1CC(NC(=O)C(N)C(C)C)C(=O)NC(C(O)=O)CC1=CC=C(O)C=C1 VBTFUDNTMCHPII-UHFFFAOYSA-N 0.000 description 2
- 201000000690 abdominal obesity-metabolic syndrome Diseases 0.000 description 2
- 230000002411 adverse Effects 0.000 description 2
- 229940035676 analgesics Drugs 0.000 description 2
- 239000005557 antagonist Substances 0.000 description 2
- 239000000730 antalgic agent Substances 0.000 description 2
- 229940124599 anti-inflammatory drug Drugs 0.000 description 2
- 230000010056 antibody-dependent cellular cytotoxicity Effects 0.000 description 2
- 230000000890 antigenic effect Effects 0.000 description 2
- 230000001640 apoptogenic effect Effects 0.000 description 2
- 229940009098 aspartate Drugs 0.000 description 2
- 229960005370 atorvastatin Drugs 0.000 description 2
- RITAVMQDGBJQJZ-FMIVXFBMSA-N axitinib Chemical compound CNC(=O)C1=CC=CC=C1SC1=CC=C(C(\C=C\C=2N=CC=CC=2)=NN2)C2=C1 RITAVMQDGBJQJZ-FMIVXFBMSA-N 0.000 description 2
- SESFRYSPDFLNCH-UHFFFAOYSA-N benzyl benzoate Chemical compound C=1C=CC=CC=1C(=O)OCC1=CC=CC=C1 SESFRYSPDFLNCH-UHFFFAOYSA-N 0.000 description 2
- 230000015572 biosynthetic process Effects 0.000 description 2
- 229960002685 biotin Drugs 0.000 description 2
- 235000020958 biotin Nutrition 0.000 description 2
- 239000011616 biotin Substances 0.000 description 2
- 239000000969 carrier Substances 0.000 description 2
- 229960002412 cediranib Drugs 0.000 description 2
- 238000006243 chemical reaction Methods 0.000 description 2
- 229940044683 chemotherapy drug Drugs 0.000 description 2
- 230000004540 complement-dependent cytotoxicity Effects 0.000 description 2
- 238000007796 conventional method Methods 0.000 description 2
- 229940111134 coxibs Drugs 0.000 description 2
- 239000003255 cyclooxygenase 2 inhibitor Substances 0.000 description 2
- 125000000151 cysteine group Chemical group N[C@@H](CS)C(=O)* 0.000 description 2
- 108010081447 cytochrophin-4 Proteins 0.000 description 2
- 239000002619 cytotoxin Substances 0.000 description 2
- 239000000839 emulsion Substances 0.000 description 2
- 238000005516 engineering process Methods 0.000 description 2
- 239000003925 fat Substances 0.000 description 2
- 238000001943 fluorescence-activated cell sorting Methods 0.000 description 2
- 229960003765 fluvastatin Drugs 0.000 description 2
- 239000000499 gel Substances 0.000 description 2
- 238000010353 genetic engineering Methods 0.000 description 2
- 108010042598 glutamyl-aspartyl-glycine Proteins 0.000 description 2
- 230000013595 glycosylation Effects 0.000 description 2
- 238000006206 glycosylation reaction Methods 0.000 description 2
- 108010048994 glycyl-tyrosyl-alanine Proteins 0.000 description 2
- 230000012010 growth Effects 0.000 description 2
- 230000013632 homeostatic process Effects 0.000 description 2
- 235000003642 hunger Nutrition 0.000 description 2
- 230000001900 immune effect Effects 0.000 description 2
- 230000028993 immune response Effects 0.000 description 2
- 230000005847 immunogenicity Effects 0.000 description 2
- 239000003018 immunosuppressive agent Substances 0.000 description 2
- 230000001771 impaired effect Effects 0.000 description 2
- 208000027866 inflammatory disease Diseases 0.000 description 2
- 238000002955 isolation Methods 0.000 description 2
- 229960000310 isoleucine Drugs 0.000 description 2
- 108010060857 isoleucyl-valyl-tyrosine Proteins 0.000 description 2
- 108010012058 leucyltyrosine Proteins 0.000 description 2
- 235000019421 lipase Nutrition 0.000 description 2
- 239000007788 liquid Substances 0.000 description 2
- 210000004185 liver Anatomy 0.000 description 2
- 108010064235 lysylglycine Proteins 0.000 description 2
- 238000009448 modified atmosphere packaging Methods 0.000 description 2
- 235000019837 monoammonium phosphate Nutrition 0.000 description 2
- 238000010172 mouse model Methods 0.000 description 2
- 210000004400 mucous membrane Anatomy 0.000 description 2
- 201000000050 myeloid neoplasm Diseases 0.000 description 2
- 239000013642 negative control Substances 0.000 description 2
- 229950008835 neratinib Drugs 0.000 description 2
- JWNPDZNEKVCWMY-VQHVLOKHSA-N neratinib Chemical compound C=12C=C(NC(=O)\C=C\CN(C)C)C(OCC)=CC2=NC=C(C#N)C=1NC(C=C1Cl)=CC=C1OCC1=CC=CC=N1 JWNPDZNEKVCWMY-VQHVLOKHSA-N 0.000 description 2
- 229910052757 nitrogen Inorganic materials 0.000 description 2
- 239000003921 oil Substances 0.000 description 2
- 230000003287 optical effect Effects 0.000 description 2
- CUIHSIWYWATEQL-UHFFFAOYSA-N pazopanib Chemical compound C1=CC2=C(C)N(C)N=C2C=C1N(C)C(N=1)=CC=NC=1NC1=CC=C(C)C(S(N)(=O)=O)=C1 CUIHSIWYWATEQL-UHFFFAOYSA-N 0.000 description 2
- 239000000546 pharmaceutical excipient Substances 0.000 description 2
- 230000000144 pharmacologic effect Effects 0.000 description 2
- 229910052698 phosphorus Inorganic materials 0.000 description 2
- BASFCYQUMIYNBI-UHFFFAOYSA-N platinum Chemical compound [Pt] BASFCYQUMIYNBI-UHFFFAOYSA-N 0.000 description 2
- 229920001223 polyethylene glycol Polymers 0.000 description 2
- 229960002965 pravastatin Drugs 0.000 description 2
- TUZYXOIXSAXUGO-PZAWKZKUSA-N pravastatin Chemical compound C1=C[C@H](C)[C@H](CC[C@@H](O)C[C@@H](O)CC(O)=O)[C@H]2[C@@H](OC(=O)[C@@H](C)CC)C[C@H](O)C=C21 TUZYXOIXSAXUGO-PZAWKZKUSA-N 0.000 description 2
- 230000002062 proliferating effect Effects 0.000 description 2
- 238000000746 purification Methods 0.000 description 2
- 229960004836 regorafenib Drugs 0.000 description 2
- FNHKPVJBJVTLMP-UHFFFAOYSA-N regorafenib Chemical compound C1=NC(C(=O)NC)=CC(OC=2C=C(F)C(NC(=O)NC=3C=C(C(Cl)=CC=3)C(F)(F)F)=CC=2)=C1 FNHKPVJBJVTLMP-UHFFFAOYSA-N 0.000 description 2
- 230000000717 retained effect Effects 0.000 description 2
- 208000004644 retinal vein occlusion Diseases 0.000 description 2
- 230000002441 reversible effect Effects 0.000 description 2
- 206010039073 rheumatoid arthritis Diseases 0.000 description 2
- 229960000672 rosuvastatin Drugs 0.000 description 2
- BPRHUIZQVSMCRT-VEUZHWNKSA-N rosuvastatin Chemical compound CC(C)C1=NC(N(C)S(C)(=O)=O)=NC(C=2C=CC(F)=CC=2)=C1\C=C\[C@@H](O)C[C@@H](O)CC(O)=O BPRHUIZQVSMCRT-VEUZHWNKSA-N 0.000 description 2
- 102200148758 rs116840795 Human genes 0.000 description 2
- 150000003839 salts Chemical class 0.000 description 2
- 238000009738 saturating Methods 0.000 description 2
- 238000013207 serial dilution Methods 0.000 description 2
- 150000003384 small molecules Chemical class 0.000 description 2
- 230000037351 starvation Effects 0.000 description 2
- 208000024891 symptom Diseases 0.000 description 2
- 230000009885 systemic effect Effects 0.000 description 2
- 108010033670 threonyl-aspartyl-tyrosine Proteins 0.000 description 2
- 210000001519 tissue Anatomy 0.000 description 2
- 108010020532 tyrosyl-proline Proteins 0.000 description 2
- 239000004474 valine Substances 0.000 description 2
- BEJKOYIMCGMNRB-GRHHLOCNSA-N (2s)-2-amino-3-(4-hydroxyphenyl)propanoic acid;(2s)-2-amino-3-phenylpropanoic acid Chemical compound OC(=O)[C@@H](N)CC1=CC=CC=C1.OC(=O)[C@@H](N)CC1=CC=C(O)C=C1 BEJKOYIMCGMNRB-GRHHLOCNSA-N 0.000 description 1
- ZQJHYRVSKHGGJY-YPKJBDGSSA-N (2s,3r)-2-[[(2s)-2-[[(2s)-1-[(2s)-2-amino-3-(4-hydroxyphenyl)propanoyl]pyrrolidine-2-carbonyl]amino]-3-phenylpropanoyl]amino]-3-hydroxybutanoic acid Chemical compound C([C@@H](C(=O)N[C@@H]([C@H](O)C)C(O)=O)NC(=O)[C@H]1N(CCC1)C(=O)[C@@H](N)CC=1C=CC(O)=CC=1)C1=CC=CC=C1 ZQJHYRVSKHGGJY-YPKJBDGSSA-N 0.000 description 1
- JRMGHBVACUJCRP-BTJKTKAUSA-N (z)-but-2-enedioic acid;4-[(4-fluoro-2-methyl-1h-indol-5-yl)oxy]-6-methoxy-7-(3-pyrrolidin-1-ylpropoxy)quinazoline Chemical compound OC(=O)\C=C/C(O)=O.COC1=CC2=C(OC=3C(=C4C=C(C)NC4=CC=3)F)N=CN=C2C=C1OCCCN1CCCC1 JRMGHBVACUJCRP-BTJKTKAUSA-N 0.000 description 1
- RZPAXNJLEKLXNO-UKNNTIGFSA-N 22-Hydroxycholesterol Chemical compound C1C=C2C[C@@H](O)CC[C@]2(C)[C@@H]2[C@@H]1[C@@H]1CC[C@H]([C@H](C)C(O)CCC(C)C)[C@@]1(C)CC2 RZPAXNJLEKLXNO-UKNNTIGFSA-N 0.000 description 1
- XXJWYDDUDKYVKI-UHFFFAOYSA-N 4-[(4-fluoro-2-methyl-1H-indol-5-yl)oxy]-6-methoxy-7-[3-(1-pyrrolidinyl)propoxy]quinazoline Chemical compound COC1=CC2=C(OC=3C(=C4C=C(C)NC4=CC=3)F)N=CN=C2C=C1OCCCN1CCCC1 XXJWYDDUDKYVKI-UHFFFAOYSA-N 0.000 description 1
- YRNWIFYIFSBPAU-UHFFFAOYSA-N 4-[4-(dimethylamino)phenyl]-n,n-dimethylaniline Chemical compound C1=CC(N(C)C)=CC=C1C1=CC=C(N(C)C)C=C1 YRNWIFYIFSBPAU-UHFFFAOYSA-N 0.000 description 1
- PVRDHKBVCDWVPD-UHFFFAOYSA-N 6,7-Dimethyl-9-(2-acetoxyethyl)isoalloxazine Chemical compound CC(=O)OCCN1C2=CC(C)=C(C)C=C2N=C2C1=NC(=O)NC2=O PVRDHKBVCDWVPD-UHFFFAOYSA-N 0.000 description 1
- VVIAGPKUTFNRDU-UHFFFAOYSA-N 6S-folinic acid Natural products C1NC=2NC(N)=NC(=O)C=2N(C=O)C1CNC1=CC=C(C(=O)NC(CCC(O)=O)C(O)=O)C=C1 VVIAGPKUTFNRDU-UHFFFAOYSA-N 0.000 description 1
- 206010069754 Acquired gene mutation Diseases 0.000 description 1
- ULXXDDBFHOBEHA-ONEGZZNKSA-N Afatinib Chemical compound N1=CN=C2C=C(OC3COCC3)C(NC(=O)/C=C/CN(C)C)=CC2=C1NC1=CC=C(F)C(Cl)=C1 ULXXDDBFHOBEHA-ONEGZZNKSA-N 0.000 description 1
- HUUOZYZWNCXTFK-INTQDDNPSA-N Ala-His-Pro Chemical compound C[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)N2CCC[C@@H]2C(=O)O)N HUUOZYZWNCXTFK-INTQDDNPSA-N 0.000 description 1
- AWNAEZICPNGAJK-FXQIFTODSA-N Ala-Met-Ser Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CCSC)C(=O)N[C@@H](CO)C(O)=O AWNAEZICPNGAJK-FXQIFTODSA-N 0.000 description 1
- LYILPUNCKACNGF-NAKRPEOUSA-N Ala-Val-Ile Chemical compound CC[C@H](C)[C@@H](C(O)=O)NC(=O)[C@H](C(C)C)NC(=O)[C@H](C)N LYILPUNCKACNGF-NAKRPEOUSA-N 0.000 description 1
- 102000002260 Alkaline Phosphatase Human genes 0.000 description 1
- 108020004774 Alkaline Phosphatase Proteins 0.000 description 1
- 206010002329 Aneurysm Diseases 0.000 description 1
- 102000006501 Angiopoietin-like Proteins Human genes 0.000 description 1
- 108010019425 Angiopoietin-like Proteins Proteins 0.000 description 1
- 101710095342 Apolipoprotein B Proteins 0.000 description 1
- 102100040202 Apolipoprotein B-100 Human genes 0.000 description 1
- 108010024284 Apolipoprotein C-II Proteins 0.000 description 1
- 102000018616 Apolipoproteins B Human genes 0.000 description 1
- 108010027006 Apolipoproteins B Proteins 0.000 description 1
- WESHVRNMNFMVBE-FXQIFTODSA-N Arg-Asn-Asp Chemical compound C(C[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H](CC(=O)O)C(=O)O)N)CN=C(N)N WESHVRNMNFMVBE-FXQIFTODSA-N 0.000 description 1
- WVNFNPGXYADPPO-BQBZGAKWSA-N Arg-Gly-Ser Chemical compound NC(N)=NCCC[C@H](N)C(=O)NCC(=O)N[C@@H](CO)C(O)=O WVNFNPGXYADPPO-BQBZGAKWSA-N 0.000 description 1
- CQMQJWRCRQSBAF-BPUTZDHNSA-N Asn-Arg-Trp Chemical compound C1=CC=C2C(=C1)C(=CN2)C[C@@H](C(=O)O)NC(=O)[C@H](CCCN=C(N)N)NC(=O)[C@H](CC(=O)N)N CQMQJWRCRQSBAF-BPUTZDHNSA-N 0.000 description 1
- JRVABKHPWDRUJF-UBHSHLNASA-N Asn-Asn-Trp Chemical compound C1=CC=C2C(=C1)C(=CN2)C[C@@H](C(=O)O)NC(=O)[C@H](CC(=O)N)NC(=O)[C@H](CC(=O)N)N JRVABKHPWDRUJF-UBHSHLNASA-N 0.000 description 1
- VLDRQOHCMKCXLY-SRVKXCTJSA-N Asn-Ser-Phe Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O VLDRQOHCMKCXLY-SRVKXCTJSA-N 0.000 description 1
- WLVLIYYBPPONRJ-GCJQMDKQSA-N Asn-Thr-Ala Chemical compound [H]N[C@@H](CC(N)=O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C)C(O)=O WLVLIYYBPPONRJ-GCJQMDKQSA-N 0.000 description 1
- WBDWQKRLTVCDSY-WHFBIAKZSA-N Asp-Gly-Asp Chemical compound OC(=O)C[C@H](N)C(=O)NCC(=O)N[C@@H](CC(O)=O)C(O)=O WBDWQKRLTVCDSY-WHFBIAKZSA-N 0.000 description 1
- OMMIEVATLAGRCK-BYPYZUCNSA-N Asp-Gly-Gly Chemical compound OC(=O)C[C@H](N)C(=O)NCC(=O)NCC(O)=O OMMIEVATLAGRCK-BYPYZUCNSA-N 0.000 description 1
- KYQNAIMCTRZLNP-QSFUFRPTSA-N Asp-Ile-Val Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](C(C)C)C(O)=O KYQNAIMCTRZLNP-QSFUFRPTSA-N 0.000 description 1
- KGHLGJAXYSVNJP-WHFBIAKZSA-N Asp-Ser-Gly Chemical compound OC(=O)C[C@H](N)C(=O)N[C@@H](CO)C(=O)NCC(O)=O KGHLGJAXYSVNJP-WHFBIAKZSA-N 0.000 description 1
- VNXQRBXEQXLERQ-CIUDSAMLSA-N Asp-Ser-Lys Chemical compound C(CCN)C[C@@H](C(=O)O)NC(=O)[C@H](CO)NC(=O)[C@H](CC(=O)O)N VNXQRBXEQXLERQ-CIUDSAMLSA-N 0.000 description 1
- USENATHVGFXRNO-SRVKXCTJSA-N Asp-Tyr-Asp Chemical compound OC(=O)C[C@H](N)C(=O)N[C@H](C(=O)N[C@@H](CC(O)=O)C(O)=O)CC1=CC=C(O)C=C1 USENATHVGFXRNO-SRVKXCTJSA-N 0.000 description 1
- DCXYFEDJOCDNAF-UHFFFAOYSA-N Asparagine Natural products OC(=O)C(N)CC(N)=O DCXYFEDJOCDNAF-UHFFFAOYSA-N 0.000 description 1
- 102100026189 Beta-galactosidase Human genes 0.000 description 1
- GAGWJHPBXLXJQN-UORFTKCHSA-N Capecitabine Chemical compound C1=C(F)C(NC(=O)OCCCCC)=NC(=O)N1[C@H]1[C@H](O)[C@H](O)[C@@H](C)O1 GAGWJHPBXLXJQN-UORFTKCHSA-N 0.000 description 1
- GAGWJHPBXLXJQN-UHFFFAOYSA-N Capecitabine Natural products C1=C(F)C(NC(=O)OCCCCC)=NC(=O)N1C1C(O)C(O)C(C)O1 GAGWJHPBXLXJQN-UHFFFAOYSA-N 0.000 description 1
- 206010007559 Cardiac failure congestive Diseases 0.000 description 1
- 229920001268 Cholestyramine Polymers 0.000 description 1
- KPSRODZRAIWAKH-JTQLQIEISA-N Ciprofibrate Natural products C1=CC(OC(C)(C)C(O)=O)=CC=C1[C@H]1C(Cl)(Cl)C1 KPSRODZRAIWAKH-JTQLQIEISA-N 0.000 description 1
- 108020004705 Codon Proteins 0.000 description 1
- 229920002911 Colestipol Polymers 0.000 description 1
- 208000035473 Communicable disease Diseases 0.000 description 1
- 241000699802 Cricetulus griseus Species 0.000 description 1
- 206010070901 Diabetic dyslipidaemia Diseases 0.000 description 1
- 206010012689 Diabetic retinopathy Diseases 0.000 description 1
- 102000004190 Enzymes Human genes 0.000 description 1
- 108090000790 Enzymes Proteins 0.000 description 1
- 241000588724 Escherichia coli Species 0.000 description 1
- 108010008177 Fd immunoglobulins Proteins 0.000 description 1
- GHASVSINZRGABV-UHFFFAOYSA-N Fluorouracil Chemical compound FC1=CNC(=O)NC1=O GHASVSINZRGABV-UHFFFAOYSA-N 0.000 description 1
- HEMJJKBWTPKOJG-UHFFFAOYSA-N Gemfibrozil Chemical compound CC1=CC=C(C)C(OCCCC(C)(C)C(O)=O)=C1 HEMJJKBWTPKOJG-UHFFFAOYSA-N 0.000 description 1
- 208000034826 Genetic Predisposition to Disease Diseases 0.000 description 1
- SHERTACNJPYHAR-ACZMJKKPSA-N Gln-Ala-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@H](C)NC(=O)[C@@H](N)CCC(N)=O SHERTACNJPYHAR-ACZMJKKPSA-N 0.000 description 1
- MGJMFSBEMSNYJL-AVGNSLFASA-N Gln-Asn-Tyr Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O MGJMFSBEMSNYJL-AVGNSLFASA-N 0.000 description 1
- CRRFJBGUGNNOCS-PEFMBERDSA-N Gln-Asp-Ile Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O CRRFJBGUGNNOCS-PEFMBERDSA-N 0.000 description 1
- LPYPANUXJGFMGV-FXQIFTODSA-N Gln-Gln-Ala Chemical compound C[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CCC(=O)N)N LPYPANUXJGFMGV-FXQIFTODSA-N 0.000 description 1
- JHPFPROFOAJRFN-IHRRRGAJSA-N Gln-Glu-Tyr Chemical compound C1=CC(=CC=C1C[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)O)NC(=O)[C@H](CCC(=O)N)N)O JHPFPROFOAJRFN-IHRRRGAJSA-N 0.000 description 1
- WLRYGVYQFXRJDA-DCAQKATOSA-N Gln-Pro-Pro Chemical compound NC(=O)CC[C@H](N)C(=O)N1CCC[C@H]1C(=O)N1[C@H](C(O)=O)CCC1 WLRYGVYQFXRJDA-DCAQKATOSA-N 0.000 description 1
- LPIKVBWNNVFHCQ-GUBZILKMSA-N Gln-Ser-Leu Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(O)=O LPIKVBWNNVFHCQ-GUBZILKMSA-N 0.000 description 1
- OTQSTOXRUBVWAP-NRPADANISA-N Gln-Ser-Val Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(O)=O OTQSTOXRUBVWAP-NRPADANISA-N 0.000 description 1
- MXOODARRORARSU-ACZMJKKPSA-N Glu-Ala-Ser Chemical compound C[C@@H](C(=O)N[C@@H](CO)C(=O)O)NC(=O)[C@H](CCC(=O)O)N MXOODARRORARSU-ACZMJKKPSA-N 0.000 description 1
- LSYFGBRDBIQYAQ-FHWLQOOXSA-N Glu-Tyr-Tyr Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O LSYFGBRDBIQYAQ-FHWLQOOXSA-N 0.000 description 1
- LZEUDRYSAZAJIO-AUTRQRHGSA-N Glu-Val-Glu Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(O)=O)C(O)=O LZEUDRYSAZAJIO-AUTRQRHGSA-N 0.000 description 1
- SOYWRINXUSUWEQ-DLOVCJGASA-N Glu-Val-Val Chemical compound CC(C)[C@@H](C(O)=O)NC(=O)[C@H](C(C)C)NC(=O)[C@@H](N)CCC(O)=O SOYWRINXUSUWEQ-DLOVCJGASA-N 0.000 description 1
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 1
- LJPIRKICOISLKN-WHFBIAKZSA-N Gly-Ala-Ser Chemical compound NCC(=O)N[C@@H](C)C(=O)N[C@@H](CO)C(O)=O LJPIRKICOISLKN-WHFBIAKZSA-N 0.000 description 1
- GNBMOZPQUXTCRW-STQMWFEESA-N Gly-Asn-Trp Chemical compound C1=CC=C2C(C[C@H](NC(=O)[C@H](CC(N)=O)NC(=O)CN)C(O)=O)=CNC2=C1 GNBMOZPQUXTCRW-STQMWFEESA-N 0.000 description 1
- RPLLQZBOVIVGMX-QWRGUYRKSA-N Gly-Asp-Phe Chemical compound [H]NCC(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O RPLLQZBOVIVGMX-QWRGUYRKSA-N 0.000 description 1
- JSLVAHYTAJJEQH-QWRGUYRKSA-N Gly-Ser-Phe Chemical compound NCC(=O)N[C@@H](CO)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 JSLVAHYTAJJEQH-QWRGUYRKSA-N 0.000 description 1
- FFALDIDGPLUDKV-ZDLURKLDSA-N Gly-Thr-Ser Chemical compound [H]NCC(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CO)C(O)=O FFALDIDGPLUDKV-ZDLURKLDSA-N 0.000 description 1
- WSWWTQYHFCBKBT-DVJZZOLTSA-N Gly-Thr-Trp Chemical compound C[C@@H](O)[C@H](NC(=O)CN)C(=O)N[C@@H](Cc1c[nH]c2ccccc12)C(O)=O WSWWTQYHFCBKBT-DVJZZOLTSA-N 0.000 description 1
- DNVDEMWIYLVIQU-RCOVLWMOSA-N Gly-Val-Asp Chemical compound NCC(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CC(O)=O)C(O)=O DNVDEMWIYLVIQU-RCOVLWMOSA-N 0.000 description 1
- 239000004471 Glycine Substances 0.000 description 1
- 102000003886 Glycoproteins Human genes 0.000 description 1
- 108090000288 Glycoproteins Proteins 0.000 description 1
- 206010019280 Heart failures Diseases 0.000 description 1
- CUEQQFOGARVNHU-VGDYDELISA-N His-Ser-Ile Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CO)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O CUEQQFOGARVNHU-VGDYDELISA-N 0.000 description 1
- FFKJUTZARGRVTH-KKUMJFAQSA-N His-Ser-Tyr Chemical compound [H]N[C@@H](CC1=CNC=N1)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O FFKJUTZARGRVTH-KKUMJFAQSA-N 0.000 description 1
- YBDOQKVAGTWZMI-XIRDDKMYSA-N His-Trp-Ser Chemical compound C1=CC=C2C(=C1)C(=CN2)C[C@@H](C(=O)N[C@@H](CO)C(=O)O)NC(=O)[C@H](CC3=CN=CN3)N YBDOQKVAGTWZMI-XIRDDKMYSA-N 0.000 description 1
- 101000935587 Homo sapiens Flavin reductase (NADPH) Proteins 0.000 description 1
- 206010020772 Hypertension Diseases 0.000 description 1
- IIXDMJNYALIKGP-DJFWLOJKSA-N Ile-Asn-His Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)N IIXDMJNYALIKGP-DJFWLOJKSA-N 0.000 description 1
- YBJWJQQBWRARLT-KBIXCLLPSA-N Ile-Gln-Ser Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CO)C(O)=O YBJWJQQBWRARLT-KBIXCLLPSA-N 0.000 description 1
- VOBYAKCXGQQFLR-LSJOCFKGSA-N Ile-Gly-Val Chemical compound CC[C@H](C)[C@H](N)C(=O)NCC(=O)N[C@@H](C(C)C)C(O)=O VOBYAKCXGQQFLR-LSJOCFKGSA-N 0.000 description 1
- PWUMCBLVWPCKNO-MGHWNKPDSA-N Ile-Leu-Tyr Chemical compound CC[C@H](C)[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 PWUMCBLVWPCKNO-MGHWNKPDSA-N 0.000 description 1
- ZLFNNVATRMCAKN-ZKWXMUAHSA-N Ile-Ser-Gly Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CO)C(=O)NCC(=O)O)N ZLFNNVATRMCAKN-ZKWXMUAHSA-N 0.000 description 1
- ZDNNDIJTUHQCAM-MXAVVETBSA-N Ile-Ser-Phe Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)O)N ZDNNDIJTUHQCAM-MXAVVETBSA-N 0.000 description 1
- MITYXXNZSZLHGG-OBAATPRFSA-N Ile-Trp-Tyr Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC1=CNC2=CC=CC=C21)C(=O)N[C@@H](CC3=CC=C(C=C3)O)C(=O)O)N MITYXXNZSZLHGG-OBAATPRFSA-N 0.000 description 1
- 102000006496 Immunoglobulin Heavy Chains Human genes 0.000 description 1
- 108010019476 Immunoglobulin Heavy Chains Proteins 0.000 description 1
- 102000013463 Immunoglobulin Light Chains Human genes 0.000 description 1
- 108010065825 Immunoglobulin Light Chains Proteins 0.000 description 1
- 108010067060 Immunoglobulin Variable Region Proteins 0.000 description 1
- 102000017727 Immunoglobulin Variable Region Human genes 0.000 description 1
- 108010065920 Insulin Lispro Proteins 0.000 description 1
- XUJNEKJLAYXESH-REOHCLBHSA-N L-Cysteine Chemical compound SC[C@H](N)C(O)=O XUJNEKJLAYXESH-REOHCLBHSA-N 0.000 description 1
- QNAYBMKLOCPYGJ-REOHCLBHSA-N L-alanine Chemical compound C[C@H](N)C(O)=O QNAYBMKLOCPYGJ-REOHCLBHSA-N 0.000 description 1
- ODKSFYDXXFIFQN-BYPYZUCNSA-N L-arginine Chemical compound OC(=O)[C@@H](N)CCCN=C(N)N ODKSFYDXXFIFQN-BYPYZUCNSA-N 0.000 description 1
- ODKSFYDXXFIFQN-BYPYZUCNSA-P L-argininium(2+) Chemical compound NC(=[NH2+])NCCC[C@H]([NH3+])C(O)=O ODKSFYDXXFIFQN-BYPYZUCNSA-P 0.000 description 1
- SHZGCJCMOBCMKK-DHVFOXMCSA-N L-fucopyranose Chemical group C[C@@H]1OC(O)[C@@H](O)[C@H](O)[C@@H]1O SHZGCJCMOBCMKK-DHVFOXMCSA-N 0.000 description 1
- HNDVDQJCIGZPNO-YFKPBYRVSA-N L-histidine Chemical compound OC(=O)[C@@H](N)CC1=CN=CN1 HNDVDQJCIGZPNO-YFKPBYRVSA-N 0.000 description 1
- FFEARJCKVFRZRR-BYPYZUCNSA-N L-methionine Chemical compound CSCC[C@H](N)C(O)=O FFEARJCKVFRZRR-BYPYZUCNSA-N 0.000 description 1
- 125000000174 L-prolyl group Chemical group [H]N1C([H])([H])C([H])([H])C([H])([H])[C@@]1([H])C(*)=O 0.000 description 1
- QIVBCDIJIAJPQS-VIFPVBQESA-N L-tryptophane Chemical compound C1=CC=C2C(C[C@H](N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-VIFPVBQESA-N 0.000 description 1
- 125000000510 L-tryptophano group Chemical group [H]C1=C([H])C([H])=C2N([H])C([H])=C(C([H])([H])[C@@]([H])(C(O[H])=O)N([H])[*])C2=C1[H] 0.000 description 1
- KZSNJWFQEVHDMF-BYPYZUCNSA-N L-valine Chemical compound CC(C)[C@H](N)C(O)=O KZSNJWFQEVHDMF-BYPYZUCNSA-N 0.000 description 1
- 239000005411 L01XE02 - Gefitinib Substances 0.000 description 1
- 239000005551 L01XE03 - Erlotinib Substances 0.000 description 1
- 239000002136 L01XE07 - Lapatinib Substances 0.000 description 1
- 239000002138 L01XE21 - Regorafenib Substances 0.000 description 1
- UIARLYUEJFELEN-LROUJFHJSA-N LSM-1231 Chemical compound C12=C3N4C5=CC=CC=C5C3=C3C(=O)NCC3=C2C2=CC=CC=C2N1[C@]1(C)[C@](CO)(O)C[C@H]4O1 UIARLYUEJFELEN-LROUJFHJSA-N 0.000 description 1
- VKOAHIRLIUESLU-ULQDDVLXSA-N Leu-Arg-Phe Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O VKOAHIRLIUESLU-ULQDDVLXSA-N 0.000 description 1
- GBDMISNMNXVTNV-XIRDDKMYSA-N Leu-Asp-Trp Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CNC2=C1C=CC=C2)C(O)=O GBDMISNMNXVTNV-XIRDDKMYSA-N 0.000 description 1
- BOFAFKVZQUMTID-AVGNSLFASA-N Leu-Gln-His Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)N BOFAFKVZQUMTID-AVGNSLFASA-N 0.000 description 1
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 description 1
- 206010024604 Lipoatrophy Diseases 0.000 description 1
- 206010049287 Lipodystrophy acquired Diseases 0.000 description 1
- 108060001084 Luciferase Proteins 0.000 description 1
- 239000005089 Luciferase Substances 0.000 description 1
- GIKFNMZSGYAPEJ-HJGDQZAQSA-N Lys-Thr-Asp Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC(O)=O)C(O)=O GIKFNMZSGYAPEJ-HJGDQZAQSA-N 0.000 description 1
- 239000004472 Lysine Substances 0.000 description 1
- 101100055876 Mus musculus Apoe gene Proteins 0.000 description 1
- YBAFDPFAUTYYRW-UHFFFAOYSA-N N-L-alpha-glutamyl-L-leucine Natural products CC(C)CC(C(O)=O)NC(=O)C(N)CCC(O)=O YBAFDPFAUTYYRW-UHFFFAOYSA-N 0.000 description 1
- ZDZOTLJHXYCWBA-VCVYQWHSSA-N N-debenzoyl-N-(tert-butoxycarbonyl)-10-deacetyltaxol Chemical compound O([C@H]1[C@H]2[C@@](C([C@H](O)C3=C(C)[C@@H](OC(=O)[C@H](O)[C@@H](NC(=O)OC(C)(C)C)C=4C=CC=CC=4)C[C@]1(O)C3(C)C)=O)(C)[C@@H](O)C[C@H]1OC[C@]12OC(=O)C)C(=O)C1=CC=CC=C1 ZDZOTLJHXYCWBA-VCVYQWHSSA-N 0.000 description 1
- 230000004988 N-glycosylation Effects 0.000 description 1
- 108091007491 NSP3 Papain-like protease domains Proteins 0.000 description 1
- 241000283973 Oryctolagus cuniculus Species 0.000 description 1
- 108090000854 Oxidoreductases Proteins 0.000 description 1
- 102000004316 Oxidoreductases Human genes 0.000 description 1
- 229930012538 Paclitaxel Natural products 0.000 description 1
- 108090000526 Papain Proteins 0.000 description 1
- 108091005804 Peptidases Proteins 0.000 description 1
- 208000018262 Peripheral vascular disease Diseases 0.000 description 1
- UHRNIXJAGGLKHP-DLOVCJGASA-N Phe-Ala-Ser Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](C)C(=O)N[C@@H](CO)C(O)=O UHRNIXJAGGLKHP-DLOVCJGASA-N 0.000 description 1
- CUMXHKAOHNWRFQ-BZSNNMDCSA-N Phe-Asp-Tyr Chemical compound C([C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(O)=O)C1=CC=CC=C1 CUMXHKAOHNWRFQ-BZSNNMDCSA-N 0.000 description 1
- WDOCBGZHAQQIBL-IHPCNDPISA-N Phe-Trp-Ser Chemical compound C([C@H](N)C(=O)N[C@@H](CC=1C2=CC=CC=C2NC=1)C(=O)N[C@@H](CO)C(O)=O)C1=CC=CC=C1 WDOCBGZHAQQIBL-IHPCNDPISA-N 0.000 description 1
- VFDRDMOMHBJGKD-UFYCRDLUSA-N Phe-Tyr-Arg Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CC2=CC=C(C=C2)O)C(=O)N[C@@H](CCCN=C(N)N)C(=O)O)N VFDRDMOMHBJGKD-UFYCRDLUSA-N 0.000 description 1
- 229920003171 Poly (ethylene oxide) Polymers 0.000 description 1
- 239000002202 Polyethylene glycol Substances 0.000 description 1
- DEDANIDYQAPTFI-IHRRRGAJSA-N Pro-Asp-Tyr Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O DEDANIDYQAPTFI-IHRRRGAJSA-N 0.000 description 1
- SPLBRAKYXGOFSO-UNQGMJICSA-N Pro-Phe-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CC1=CC=CC=C1)NC(=O)[C@@H]2CCCN2)O SPLBRAKYXGOFSO-UNQGMJICSA-N 0.000 description 1
- QKWYXRPICJEQAJ-KJEVXHAQSA-N Pro-Tyr-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CC1=CC=C(C=C1)O)NC(=O)[C@@H]2CCCN2)O QKWYXRPICJEQAJ-KJEVXHAQSA-N 0.000 description 1
- 102000004022 Protein-Tyrosine Kinases Human genes 0.000 description 1
- 108090000412 Protein-Tyrosine Kinases Proteins 0.000 description 1
- 201000004681 Psoriasis Diseases 0.000 description 1
- 206010038933 Retinopathy of prematurity Diseases 0.000 description 1
- 102100037486 Reverse transcriptase/ribonuclease H Human genes 0.000 description 1
- 241000283984 Rodentia Species 0.000 description 1
- 101150071661 SLC25A20 gene Proteins 0.000 description 1
- CTLVSHXLRVEILB-UBHSHLNASA-N Ser-Asn-Trp Chemical compound C1=CC=C2C(=C1)C(=CN2)C[C@@H](C(=O)O)NC(=O)[C@H](CC(=O)N)NC(=O)[C@H](CO)N CTLVSHXLRVEILB-UBHSHLNASA-N 0.000 description 1
- HEQPKICPPDOSIN-SRVKXCTJSA-N Ser-Asp-Tyr Chemical compound OC[C@H](N)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 HEQPKICPPDOSIN-SRVKXCTJSA-N 0.000 description 1
- UQFYNFTYDHUIMI-WHFBIAKZSA-N Ser-Gly-Ala Chemical compound OC(=O)[C@H](C)NC(=O)CNC(=O)[C@@H](N)CO UQFYNFTYDHUIMI-WHFBIAKZSA-N 0.000 description 1
- MUARUIBTKQJKFY-WHFBIAKZSA-N Ser-Gly-Asp Chemical compound [H]N[C@@H](CO)C(=O)NCC(=O)N[C@@H](CC(O)=O)C(O)=O MUARUIBTKQJKFY-WHFBIAKZSA-N 0.000 description 1
- BEAFYHFQTOTVFS-VGDYDELISA-N Ser-Ile-His Chemical compound CC[C@H](C)[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)NC(=O)[C@H](CO)N BEAFYHFQTOTVFS-VGDYDELISA-N 0.000 description 1
- FKYWFUYPVKLJLP-DCAQKATOSA-N Ser-Pro-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@@H]1CCCN1C(=O)[C@@H](N)CO FKYWFUYPVKLJLP-DCAQKATOSA-N 0.000 description 1
- WLJPJRGQRNCIQS-ZLUOBGJFSA-N Ser-Ser-Asn Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(N)=O)C(O)=O WLJPJRGQRNCIQS-ZLUOBGJFSA-N 0.000 description 1
- PQEQXWRVHQAAKS-SRVKXCTJSA-N Ser-Tyr-Asn Chemical compound NC(=O)C[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)[C@H](CO)N)CC1=CC=C(O)C=C1 PQEQXWRVHQAAKS-SRVKXCTJSA-N 0.000 description 1
- SIEBDTCABMZCLF-XGEHTFHBSA-N Ser-Val-Thr Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O SIEBDTCABMZCLF-XGEHTFHBSA-N 0.000 description 1
- MTCFGRXMJLQNBG-UHFFFAOYSA-N Serine Natural products OCC(N)C(O)=O MTCFGRXMJLQNBG-UHFFFAOYSA-N 0.000 description 1
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 1
- 208000006011 Stroke Diseases 0.000 description 1
- NINIDFKCEFEMDL-UHFFFAOYSA-N Sulfur Chemical compound [S] NINIDFKCEFEMDL-UHFFFAOYSA-N 0.000 description 1
- 210000001744 T-lymphocyte Anatomy 0.000 description 1
- NDZYTIMDOZMECO-SHGPDSBTSA-N Thr-Thr-Ala Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C)C(O)=O NDZYTIMDOZMECO-SHGPDSBTSA-N 0.000 description 1
- AYFVYJQAPQTCCC-UHFFFAOYSA-N Threonine Natural products CC(O)C(N)C(O)=O AYFVYJQAPQTCCC-UHFFFAOYSA-N 0.000 description 1
- 239000004473 Threonine Substances 0.000 description 1
- QIVBCDIJIAJPQS-UHFFFAOYSA-N Tryptophan Natural products C1=CC=C2C(CC(N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-UHFFFAOYSA-N 0.000 description 1
- ZNFPUOSTMUMUDR-JRQIVUDYSA-N Tyr-Asn-Thr Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O ZNFPUOSTMUMUDR-JRQIVUDYSA-N 0.000 description 1
- MNMYOSZWCKYEDI-JRQIVUDYSA-N Tyr-Asp-Thr Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O MNMYOSZWCKYEDI-JRQIVUDYSA-N 0.000 description 1
- AZGZDDNKFFUDEH-QWRGUYRKSA-N Tyr-Gly-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)CNC(=O)[C@@H](N)CC1=CC=C(O)C=C1 AZGZDDNKFFUDEH-QWRGUYRKSA-N 0.000 description 1
- BXPOOVDVGWEXDU-WZLNRYEVSA-N Tyr-Ile-Thr Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H]([C@@H](C)O)C(O)=O BXPOOVDVGWEXDU-WZLNRYEVSA-N 0.000 description 1
- UMSZZGTXGKHTFJ-SRVKXCTJSA-N Tyr-Ser-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CC1=CC=C(O)C=C1 UMSZZGTXGKHTFJ-SRVKXCTJSA-N 0.000 description 1
- KSGKJSFPWSMJHK-JNPHEJMOSA-N Tyr-Tyr-Thr Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H]([C@@H](C)O)C(O)=O KSGKJSFPWSMJHK-JNPHEJMOSA-N 0.000 description 1
- 108091008605 VEGF receptors Proteins 0.000 description 1
- OVLIFGQSBSNGHY-KKHAAJSZSA-N Val-Asp-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CC(=O)O)NC(=O)[C@H](C(C)C)N)O OVLIFGQSBSNGHY-KKHAAJSZSA-N 0.000 description 1
- GUIYPEKUEMQBIK-JSGCOSHPSA-N Val-Tyr-Gly Chemical compound CC(C)[C@H](N)C(=O)N[C@@H](Cc1ccc(O)cc1)C(=O)NCC(O)=O GUIYPEKUEMQBIK-JSGCOSHPSA-N 0.000 description 1
- KZSNJWFQEVHDMF-UHFFFAOYSA-N Valine Natural products CC(C)C(N)C(O)=O KZSNJWFQEVHDMF-UHFFFAOYSA-N 0.000 description 1
- 108010073929 Vascular Endothelial Growth Factor A Proteins 0.000 description 1
- 102000009484 Vascular Endothelial Growth Factor Receptors Human genes 0.000 description 1
- 241000700605 Viruses Species 0.000 description 1
- HMNZFMSWFCAGGW-XPWSMXQVSA-N [3-[hydroxy(2-hydroxyethoxy)phosphoryl]oxy-2-[(e)-octadec-9-enoyl]oxypropyl] (e)-octadec-9-enoate Chemical compound CCCCCCCC\C=C\CCCCCCCC(=O)OCC(COP(O)(=O)OCCO)OC(=O)CCCCCCC\C=C\CCCCCCCC HMNZFMSWFCAGGW-XPWSMXQVSA-N 0.000 description 1
- 230000002745 absorbent Effects 0.000 description 1
- 239000002250 absorbent Substances 0.000 description 1
- 230000002378 acidificating effect Effects 0.000 description 1
- 150000007513 acids Chemical class 0.000 description 1
- 230000009471 action Effects 0.000 description 1
- 239000004480 active ingredient Substances 0.000 description 1
- 239000013543 active substance Substances 0.000 description 1
- 239000002671 adjuvant Substances 0.000 description 1
- 229960001686 afatinib Drugs 0.000 description 1
- ULXXDDBFHOBEHA-CWDCEQMOSA-N afatinib Chemical compound N1=CN=C2C=C(O[C@@H]3COCC3)C(NC(=O)/C=C/CN(C)C)=CC2=C1NC1=CC=C(F)C(Cl)=C1 ULXXDDBFHOBEHA-CWDCEQMOSA-N 0.000 description 1
- 206010064930 age-related macular degeneration Diseases 0.000 description 1
- 230000001270 agonistic effect Effects 0.000 description 1
- 235000004279 alanine Nutrition 0.000 description 1
- 238000012867 alanine scanning Methods 0.000 description 1
- 125000001931 aliphatic group Chemical group 0.000 description 1
- 230000001668 ameliorated effect Effects 0.000 description 1
- 150000001408 amides Chemical class 0.000 description 1
- 125000003277 amino group Chemical group 0.000 description 1
- 239000003708 ampul Substances 0.000 description 1
- 230000002491 angiogenic effect Effects 0.000 description 1
- 125000000129 anionic group Chemical group 0.000 description 1
- 230000003042 antagnostic effect Effects 0.000 description 1
- 230000009830 antibody antigen interaction Effects 0.000 description 1
- 239000003524 antilipemic agent Substances 0.000 description 1
- 239000003080 antimitotic agent Substances 0.000 description 1
- 238000013459 approach Methods 0.000 description 1
- ODKSFYDXXFIFQN-UHFFFAOYSA-N arginine Natural products OC(=O)C(N)CCCNC(N)=N ODKSFYDXXFIFQN-UHFFFAOYSA-N 0.000 description 1
- 108010084758 arginyl-tyrosyl-aspartic acid Proteins 0.000 description 1
- 206010003246 arthritis Diseases 0.000 description 1
- 125000003118 aryl group Chemical group 0.000 description 1
- 229960001230 asparagine Drugs 0.000 description 1
- 235000009582 asparagine Nutrition 0.000 description 1
- 108010093581 aspartyl-proline Proteins 0.000 description 1
- 229940120638 avastin Drugs 0.000 description 1
- 229960003005 axitinib Drugs 0.000 description 1
- VSRXQHXAPYXROS-UHFFFAOYSA-N azanide;cyclobutane-1,1-dicarboxylic acid;platinum(2+) Chemical compound [NH2-].[NH2-].[Pt+2].OC(=O)C1(C(O)=O)CCC1 VSRXQHXAPYXROS-UHFFFAOYSA-N 0.000 description 1
- 230000001580 bacterial effect Effects 0.000 description 1
- 229960002903 benzyl benzoate Drugs 0.000 description 1
- 108010005774 beta-Galactosidase Proteins 0.000 description 1
- 229960000397 bevacizumab Drugs 0.000 description 1
- 229960000516 bezafibrate Drugs 0.000 description 1
- IIBYAHWJQTYFKB-UHFFFAOYSA-N bezafibrate Chemical compound C1=CC(OC(C)(C)C(O)=O)=CC=C1CCNC(=O)C1=CC=C(Cl)C=C1 IIBYAHWJQTYFKB-UHFFFAOYSA-N 0.000 description 1
- 229920000080 bile acid sequestrant Polymers 0.000 description 1
- 229940096699 bile acid sequestrants Drugs 0.000 description 1
- 239000003124 biologic agent Substances 0.000 description 1
- 230000033228 biological regulation Effects 0.000 description 1
- 230000005540 biological transmission Effects 0.000 description 1
- 210000001124 body fluid Anatomy 0.000 description 1
- 239000010839 body fluid Substances 0.000 description 1
- 102220349284 c.287A>T Human genes 0.000 description 1
- 101150102633 cact gene Proteins 0.000 description 1
- 229960004117 capecitabine Drugs 0.000 description 1
- 239000002775 capsule Substances 0.000 description 1
- 229960004562 carboplatin Drugs 0.000 description 1
- 150000001735 carboxylic acids Chemical class 0.000 description 1
- 239000004359 castor oil Substances 0.000 description 1
- 235000019438 castor oil Nutrition 0.000 description 1
- 125000002091 cationic group Chemical group 0.000 description 1
- 201000005667 central retinal vein occlusion Diseases 0.000 description 1
- 208000026106 cerebrovascular disease Diseases 0.000 description 1
- 229960005110 cerivastatin Drugs 0.000 description 1
- SEERZIQQUAZTOL-ANMDKAQQSA-N cerivastatin Chemical compound COCC1=C(C(C)C)N=C(C(C)C)C(\C=C\[C@@H](O)C[C@@H](O)CC(O)=O)=C1C1=CC=C(F)C=C1 SEERZIQQUAZTOL-ANMDKAQQSA-N 0.000 description 1
- 238000012412 chemical coupling Methods 0.000 description 1
- 238000007385 chemical modification Methods 0.000 description 1
- 230000000973 chemotherapeutic effect Effects 0.000 description 1
- 210000004978 chinese hamster ovary cell Anatomy 0.000 description 1
- 229960002174 ciprofibrate Drugs 0.000 description 1
- KPSRODZRAIWAKH-UHFFFAOYSA-N ciprofibrate Chemical compound C1=CC(OC(C)(C)C(O)=O)=CC=C1C1C(Cl)(Cl)C1 KPSRODZRAIWAKH-UHFFFAOYSA-N 0.000 description 1
- 229960004316 cisplatin Drugs 0.000 description 1
- DQLATGHUWYMOKM-UHFFFAOYSA-L cisplatin Chemical compound N[Pt](N)(Cl)Cl DQLATGHUWYMOKM-UHFFFAOYSA-L 0.000 description 1
- 231100000313 clinical toxicology Toxicity 0.000 description 1
- 229960001214 clofibrate Drugs 0.000 description 1
- KNHUKKLJHYUCFP-UHFFFAOYSA-N clofibrate Chemical compound CCOC(=O)C(C)(C)OC1=CC=C(Cl)C=C1 KNHUKKLJHYUCFP-UHFFFAOYSA-N 0.000 description 1
- 229960002604 colestipol Drugs 0.000 description 1
- GMRWGQCZJGVHKL-UHFFFAOYSA-N colestipol Chemical compound ClCC1CO1.NCCNCCNCCNCCN GMRWGQCZJGVHKL-UHFFFAOYSA-N 0.000 description 1
- 238000012875 competitive assay Methods 0.000 description 1
- 230000000295 complement effect Effects 0.000 description 1
- 238000013329 compounding Methods 0.000 description 1
- 238000004590 computer program Methods 0.000 description 1
- 208000029078 coronary artery disease Diseases 0.000 description 1
- 239000013078 crystal Substances 0.000 description 1
- 238000012258 culturing Methods 0.000 description 1
- XUJNEKJLAYXESH-UHFFFAOYSA-N cysteine Natural products SCC(N)C(O)=O XUJNEKJLAYXESH-UHFFFAOYSA-N 0.000 description 1
- 235000018417 cysteine Nutrition 0.000 description 1
- 230000006378 damage Effects 0.000 description 1
- CYQFCXCEBYINGO-IAGOWNOFSA-N delta1-THC Chemical class C1=C(C)CC[C@H]2C(C)(C)OC3=CC(CCCCC)=CC(O)=C3[C@@H]21 CYQFCXCEBYINGO-IAGOWNOFSA-N 0.000 description 1
- 230000001419 dependent effect Effects 0.000 description 1
- 238000000502 dialysis Methods 0.000 description 1
- 235000005911 diet Nutrition 0.000 description 1
- 230000037213 diet Effects 0.000 description 1
- 235000014113 dietary fatty acids Nutrition 0.000 description 1
- 230000029087 digestion Effects 0.000 description 1
- 239000000539 dimer Substances 0.000 description 1
- 208000037765 diseases and disorders Diseases 0.000 description 1
- 229960003668 docetaxel Drugs 0.000 description 1
- 231100000673 dose–response relationship Toxicity 0.000 description 1
- 239000012636 effector Substances 0.000 description 1
- 230000008030 elimination Effects 0.000 description 1
- 238000003379 elimination reaction Methods 0.000 description 1
- 210000001671 embryonic stem cell Anatomy 0.000 description 1
- 230000001804 emulsifying effect Effects 0.000 description 1
- 230000012202 endocytosis Effects 0.000 description 1
- 229940088598 enzyme Drugs 0.000 description 1
- 229960001433 erlotinib Drugs 0.000 description 1
- AAKJLRGGTJKAMG-UHFFFAOYSA-N erlotinib Chemical compound C=12C=C(OCCOC)C(OCCOC)=CC2=NC=NC=1NC1=CC=CC(C#C)=C1 AAKJLRGGTJKAMG-UHFFFAOYSA-N 0.000 description 1
- DTMGIJFHGGCSLO-FIAQIACWSA-N ethyl (4z,7z,10z,13z,16z,19z)-docosa-4,7,10,13,16,19-hexaenoate;ethyl (5z,8z,11z,14z,17z)-icosa-5,8,11,14,17-pentaenoate Chemical compound CCOC(=O)CCC\C=C/C\C=C/C\C=C/C\C=C/C\C=C/CC.CCOC(=O)CC\C=C/C\C=C/C\C=C/C\C=C/C\C=C/C\C=C/CC DTMGIJFHGGCSLO-FIAQIACWSA-N 0.000 description 1
- 238000011156 evaluation Methods 0.000 description 1
- 230000005284 excitation Effects 0.000 description 1
- 230000001747 exhibiting effect Effects 0.000 description 1
- OLNTVTPDXPETLC-XPWALMASSA-N ezetimibe Chemical compound N1([C@@H]([C@H](C1=O)CC[C@H](O)C=1C=CC(F)=CC=1)C=1C=CC(O)=CC=1)C1=CC=C(F)C=C1 OLNTVTPDXPETLC-XPWALMASSA-N 0.000 description 1
- 229960000815 ezetimibe Drugs 0.000 description 1
- 201000011110 familial lipoprotein lipase deficiency Diseases 0.000 description 1
- 235000019197 fats Nutrition 0.000 description 1
- 229930195729 fatty acid Natural products 0.000 description 1
- 239000000194 fatty acid Substances 0.000 description 1
- 150000004665 fatty acids Chemical class 0.000 description 1
- 230000001723 fibrinogenic effect Effects 0.000 description 1
- 238000000684 flow cytometry Methods 0.000 description 1
- MHMNJMPURVTYEJ-UHFFFAOYSA-N fluorescein-5-isothiocyanate Chemical compound O1C(=O)C2=CC(N=C=S)=CC=C2C21C1=CC=C(O)C=C1OC1=CC(O)=CC=C21 MHMNJMPURVTYEJ-UHFFFAOYSA-N 0.000 description 1
- 229960002949 fluorouracil Drugs 0.000 description 1
- VVIAGPKUTFNRDU-ABLWVSNPSA-N folinic acid Chemical compound C1NC=2NC(N)=NC(=O)C=2N(C=O)C1CNC1=CC=C(C(=O)N[C@@H](CCC(O)=O)C(O)=O)C=C1 VVIAGPKUTFNRDU-ABLWVSNPSA-N 0.000 description 1
- 235000008191 folinic acid Nutrition 0.000 description 1
- 239000011672 folinic acid Substances 0.000 description 1
- 230000004927 fusion Effects 0.000 description 1
- 108020001507 fusion proteins Proteins 0.000 description 1
- 102000037865 fusion proteins Human genes 0.000 description 1
- 229960002584 gefitinib Drugs 0.000 description 1
- XGALLCVXEZPNRQ-UHFFFAOYSA-N gefitinib Chemical compound C=12C=C(OCCCN3CCOCC3)C(OC)=CC2=NC=NC=1NC1=CC=C(F)C(Cl)=C1 XGALLCVXEZPNRQ-UHFFFAOYSA-N 0.000 description 1
- SDUQYLNIPVEERB-QPPQHZFASA-N gemcitabine Chemical compound O=C1N=C(N)C=CN1[C@H]1C(F)(F)[C@H](O)[C@@H](CO)O1 SDUQYLNIPVEERB-QPPQHZFASA-N 0.000 description 1
- 229960005277 gemcitabine Drugs 0.000 description 1
- 229960003627 gemfibrozil Drugs 0.000 description 1
- 238000007429 general method Methods 0.000 description 1
- 239000003862 glucocorticoid Substances 0.000 description 1
- 239000008103 glucose Substances 0.000 description 1
- 229930195712 glutamate Natural products 0.000 description 1
- ZDXPYRJPNDTMRX-UHFFFAOYSA-N glutamine Natural products OC(=O)C(N)CCC(N)=O ZDXPYRJPNDTMRX-UHFFFAOYSA-N 0.000 description 1
- ZEMPKEQAKRGZGQ-XOQCFJPHSA-N glycerol triricinoleate Natural products CCCCCC[C@@H](O)CC=CCCCCCCCC(=O)OC[C@@H](COC(=O)CCCCCCCC=CC[C@@H](O)CCCCCC)OC(=O)CCCCCCCC=CC[C@H](O)CCCCCC ZEMPKEQAKRGZGQ-XOQCFJPHSA-N 0.000 description 1
- 108010074027 glycyl-seryl-phenylalanine Proteins 0.000 description 1
- 230000036541 health Effects 0.000 description 1
- 239000000833 heterodimer Substances 0.000 description 1
- 108010028295 histidylhistidine Proteins 0.000 description 1
- 239000000710 homodimer Substances 0.000 description 1
- 229940038661 humalog Drugs 0.000 description 1
- 102000053580 human ANGPTL3 Human genes 0.000 description 1
- 238000011577 humanized mouse model Methods 0.000 description 1
- 238000009396 hybridization Methods 0.000 description 1
- 230000007062 hydrolysis Effects 0.000 description 1
- 238000006460 hydrolysis reaction Methods 0.000 description 1
- 238000000099 in vitro assay Methods 0.000 description 1
- 238000005462 in vivo assay Methods 0.000 description 1
- 238000010348 incorporation Methods 0.000 description 1
- 238000011534 incubation Methods 0.000 description 1
- 208000015181 infectious disease Diseases 0.000 description 1
- 210000000936 intestine Anatomy 0.000 description 1
- 238000007918 intramuscular administration Methods 0.000 description 1
- 238000010255 intramuscular injection Methods 0.000 description 1
- 239000007927 intramuscular injection Substances 0.000 description 1
- 238000007912 intraperitoneal administration Methods 0.000 description 1
- 238000010253 intravenous injection Methods 0.000 description 1
- 229950010897 iproplatin Drugs 0.000 description 1
- 229960004768 irinotecan Drugs 0.000 description 1
- UWKQSNNFCGGAFS-XIFFEERXSA-N irinotecan Chemical compound C1=C2C(CC)=C3CN(C(C4=C([C@@](C(=O)OC4)(O)CC)C=4)=O)C=4C3=NC2=CC=C1OC(=O)N(CC1)CCC1N1CCCCC1 UWKQSNNFCGGAFS-XIFFEERXSA-N 0.000 description 1
- 230000001788 irregular Effects 0.000 description 1
- AGPKZVBTJJNPAG-UHFFFAOYSA-N isoleucine Natural products CCC(C)C(N)C(O)=O AGPKZVBTJJNPAG-UHFFFAOYSA-N 0.000 description 1
- 239000000644 isotonic solution Substances 0.000 description 1
- 229960004891 lapatinib Drugs 0.000 description 1
- BCFGMOOMADDAQU-UHFFFAOYSA-N lapatinib Chemical compound O1C(CNCCS(=O)(=O)C)=CC=C1C1=CC=C(N=CN=C2NC=3C=C(Cl)C(OCC=4C=C(F)C=CC=4)=CC=3)C2=C1 BCFGMOOMADDAQU-UHFFFAOYSA-N 0.000 description 1
- 229960001691 leucovorin Drugs 0.000 description 1
- 208000006132 lipodystrophy Diseases 0.000 description 1
- 230000008604 lipoprotein metabolism Effects 0.000 description 1
- 239000007791 liquid phase Substances 0.000 description 1
- 208000002780 macular degeneration Diseases 0.000 description 1
- 238000012423 maintenance Methods 0.000 description 1
- 210000004962 mammalian cell Anatomy 0.000 description 1
- 238000013507 mapping Methods 0.000 description 1
- 238000005259 measurement Methods 0.000 description 1
- 229940126601 medicinal product Drugs 0.000 description 1
- 239000012528 membrane Substances 0.000 description 1
- 210000004379 membrane Anatomy 0.000 description 1
- 230000004060 metabolic process Effects 0.000 description 1
- 239000002207 metabolite Substances 0.000 description 1
- 229930182817 methionine Natural products 0.000 description 1
- 239000003094 microcapsule Substances 0.000 description 1
- 239000011859 microparticle Substances 0.000 description 1
- 230000003278 mimic effect Effects 0.000 description 1
- 238000000302 molecular modelling Methods 0.000 description 1
- 239000000178 monomer Substances 0.000 description 1
- 210000000214 mouth Anatomy 0.000 description 1
- 238000002703 mutagenesis Methods 0.000 description 1
- 231100000350 mutagenesis Toxicity 0.000 description 1
- LBWFXVZLPYTWQI-IPOVEDGCSA-N n-[2-(diethylamino)ethyl]-5-[(z)-(5-fluoro-2-oxo-1h-indol-3-ylidene)methyl]-2,4-dimethyl-1h-pyrrole-3-carboxamide;(2s)-2-hydroxybutanedioic acid Chemical compound OC(=O)[C@@H](O)CC(O)=O.CCN(CC)CCNC(=O)C1=C(C)NC(\C=C/2C3=CC(F)=CC=C3NC\2=O)=C1C LBWFXVZLPYTWQI-IPOVEDGCSA-N 0.000 description 1
- 230000001613 neoplastic effect Effects 0.000 description 1
- 230000003472 neutralizing effect Effects 0.000 description 1
- 229940080607 nexavar Drugs 0.000 description 1
- 239000002736 nonionic surfactant Substances 0.000 description 1
- 235000019198 oils Nutrition 0.000 description 1
- 239000002674 ointment Substances 0.000 description 1
- 238000006384 oligomerization reaction Methods 0.000 description 1
- 230000002018 overexpression Effects 0.000 description 1
- 229960001756 oxaliplatin Drugs 0.000 description 1
- DWAFYCQODLXJNR-BNTLRKBRSA-L oxaliplatin Chemical compound O1C(=O)C(=O)O[Pt]11N[C@@H]2CCCC[C@H]2N1 DWAFYCQODLXJNR-BNTLRKBRSA-L 0.000 description 1
- 229960001592 paclitaxel Drugs 0.000 description 1
- 229940090244 palladia Drugs 0.000 description 1
- 229940055729 papain Drugs 0.000 description 1
- 235000019834 papain Nutrition 0.000 description 1
- 230000036961 partial effect Effects 0.000 description 1
- 239000002245 particle Substances 0.000 description 1
- 239000006072 paste Substances 0.000 description 1
- 239000013610 patient sample Substances 0.000 description 1
- 229960000639 pazopanib Drugs 0.000 description 1
- 230000007030 peptide scission Effects 0.000 description 1
- 238000002823 phage display Methods 0.000 description 1
- 239000012071 phase Substances 0.000 description 1
- WVDDGKGOMKODPV-ZQBYOMGUSA-N phenyl(114C)methanol Chemical compound O[14CH2]C1=CC=CC=C1 WVDDGKGOMKODPV-ZQBYOMGUSA-N 0.000 description 1
- COLNVLDHVKWLRT-UHFFFAOYSA-N phenylalanine Natural products OC(=O)C(N)CC1=CC=CC=C1 COLNVLDHVKWLRT-UHFFFAOYSA-N 0.000 description 1
- 108010073101 phenylalanylleucine Proteins 0.000 description 1
- 230000004962 physiological condition Effects 0.000 description 1
- 239000006187 pill Substances 0.000 description 1
- 229960002797 pitavastatin Drugs 0.000 description 1
- VGYFMXBACGZSIL-MCBHFWOFSA-N pitavastatin Chemical compound OC(=O)C[C@H](O)C[C@H](O)\C=C\C1=C(C2CC2)N=C2C=CC=CC2=C1C1=CC=C(F)C=C1 VGYFMXBACGZSIL-MCBHFWOFSA-N 0.000 description 1
- 229940096701 plain lipid modifying drug hmg coa reductase inhibitors Drugs 0.000 description 1
- 229910052697 platinum Inorganic materials 0.000 description 1
- 239000000244 polyoxyethylene sorbitan monooleate Substances 0.000 description 1
- 235000010482 polyoxyethylene sorbitan monooleate Nutrition 0.000 description 1
- 229920000053 polysorbate 80 Polymers 0.000 description 1
- 229940068968 polysorbate 80 Drugs 0.000 description 1
- 239000000843 powder Substances 0.000 description 1
- 238000012545 processing Methods 0.000 description 1
- 230000002035 prolonged effect Effects 0.000 description 1
- 108010087846 prolyl-prolyl-glycine Proteins 0.000 description 1
- 235000019419 proteases Nutrition 0.000 description 1
- 238000000159 protein binding assay Methods 0.000 description 1
- 230000006337 proteolytic cleavage Effects 0.000 description 1
- QHGVXILFMXYDRS-UHFFFAOYSA-N pyraclofos Chemical compound C1=C(OP(=O)(OCC)SCCC)C=NN1C1=CC=C(Cl)C=C1 QHGVXILFMXYDRS-UHFFFAOYSA-N 0.000 description 1
- 238000010223 real-time analysis Methods 0.000 description 1
- 108091006082 receptor inhibitors Proteins 0.000 description 1
- 102000005962 receptors Human genes 0.000 description 1
- 108020003175 receptors Proteins 0.000 description 1
- 238000003259 recombinant expression Methods 0.000 description 1
- 210000000664 rectum Anatomy 0.000 description 1
- 238000004153 renaturation Methods 0.000 description 1
- 238000011160 research Methods 0.000 description 1
- 230000004044 response Effects 0.000 description 1
- PYWVYCXTNDRMGF-UHFFFAOYSA-N rhodamine B Chemical compound [Cl-].C=12C=CC(=[N+](CC)CC)C=C2OC2=CC(N(CC)CC)=CC=C2C=1C1=CC=CC=C1C(O)=O PYWVYCXTNDRMGF-UHFFFAOYSA-N 0.000 description 1
- 102220210869 rs1057524586 Human genes 0.000 description 1
- 102220220520 rs1060503090 Human genes 0.000 description 1
- 102220206698 rs142514490 Human genes 0.000 description 1
- 102220325921 rs1555376589 Human genes 0.000 description 1
- 102220142694 rs192332456 Human genes 0.000 description 1
- 102200164344 rs63751661 Human genes 0.000 description 1
- 238000003118 sandwich ELISA Methods 0.000 description 1
- 238000012216 screening Methods 0.000 description 1
- 230000028327 secretion Effects 0.000 description 1
- 239000008159 sesame oil Substances 0.000 description 1
- 235000011803 sesame oil Nutrition 0.000 description 1
- 239000011780 sodium chloride Substances 0.000 description 1
- 239000007790 solid phase Substances 0.000 description 1
- 230000037439 somatic mutation Effects 0.000 description 1
- 229960003787 sorafenib Drugs 0.000 description 1
- 239000003549 soybean oil Substances 0.000 description 1
- 235000012424 soybean oil Nutrition 0.000 description 1
- 210000000952 spleen Anatomy 0.000 description 1
- 238000010561 standard procedure Methods 0.000 description 1
- 239000000758 substrate Substances 0.000 description 1
- 150000005846 sugar alcohols Polymers 0.000 description 1
- 125000000472 sulfonyl group Chemical group *S(*)(=O)=O 0.000 description 1
- 229910052717 sulfur Inorganic materials 0.000 description 1
- 239000011593 sulfur Substances 0.000 description 1
- 229960001796 sunitinib Drugs 0.000 description 1
- WINHZLLDWRZWRT-ATVHPVEESA-N sunitinib Chemical compound CCN(CC)CCNC(=O)C1=C(C)NC(\C=C/2C3=CC(F)=CC=C3NC\2=O)=C1C WINHZLLDWRZWRT-ATVHPVEESA-N 0.000 description 1
- 239000000829 suppository Substances 0.000 description 1
- 230000001629 suppression Effects 0.000 description 1
- 229940034785 sutent Drugs 0.000 description 1
- 230000002195 synergetic effect Effects 0.000 description 1
- 239000003826 tablet Substances 0.000 description 1
- RCINICONZNJXQF-MZXODVADSA-N taxol Chemical compound O([C@@H]1[C@@]2(C[C@@H](C(C)=C(C2(C)C)[C@H](C([C@]2(C)[C@@H](O)C[C@H]3OC[C@]3([C@H]21)OC(C)=O)=O)OC(=O)C)OC(=O)[C@H](O)[C@@H](NC(=O)C=1C=CC=CC=1)C=1C=CC=CC=1)O)C(=O)C1=CC=CC=C1 RCINICONZNJXQF-MZXODVADSA-N 0.000 description 1
- 229960005048 toceranib Drugs 0.000 description 1
- 206010044008 tonsillitis Diseases 0.000 description 1
- 238000012546 transfer Methods 0.000 description 1
- 230000032258 transport Effects 0.000 description 1
- UFTFJSFQGQCHQW-UHFFFAOYSA-N triformin Chemical compound O=COCC(OC=O)COC=O UFTFJSFQGQCHQW-UHFFFAOYSA-N 0.000 description 1
- 108010080629 tryptophan-leucine Proteins 0.000 description 1
- 208000001072 type 2 diabetes mellitus Diseases 0.000 description 1
- OUYCCCASQSFEME-UHFFFAOYSA-N tyrosine Natural products OC(=O)C(N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-UHFFFAOYSA-N 0.000 description 1
- 239000013598 vector Substances 0.000 description 1
- 210000003462 vein Anatomy 0.000 description 1
- 229940069559 votrient Drugs 0.000 description 1
- 239000011534 wash buffer Substances 0.000 description 1
- 238000005406 washing Methods 0.000 description 1
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 1
- 239000001993 wax Substances 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/26—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against hormones ; against hormone releasing or inhibiting factors
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K31/00—Medicinal preparations containing organic active ingredients
- A61K31/21—Esters, e.g. nitroglycerine, selenocyanates
- A61K31/215—Esters, e.g. nitroglycerine, selenocyanates of carboxylic acids
- A61K31/216—Esters, e.g. nitroglycerine, selenocyanates of carboxylic acids of acids having aromatic rings, e.g. benactizyne, clofibrate
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K38/00—Medicinal preparations containing peptides
- A61K38/16—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- A61K38/17—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K38/00—Medicinal preparations containing peptides
- A61K38/16—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- A61K38/17—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- A61K38/18—Growth factors; Growth regulators
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/395—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/395—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum
- A61K39/39533—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum against materials from animals
- A61K39/3955—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum against materials from animals against proteinaceous materials, e.g. enzymes, hormones, lymphokines
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K45/00—Medicinal preparations containing active ingredients not provided for in groups A61K31/00 - A61K41/00
- A61K45/06—Mixtures of active ingredients without chemical characterisation, e.g. antiphlogistics and cardiaca
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K48/00—Medicinal preparations containing genetic material which is inserted into cells of the living body to treat genetic diseases; Gene therapy
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K49/00—Preparations for testing in vivo
- A61K49/06—Nuclear magnetic resonance [NMR] contrast preparations; Magnetic resonance imaging [MRI] contrast preparations
- A61K49/08—Nuclear magnetic resonance [NMR] contrast preparations; Magnetic resonance imaging [MRI] contrast preparations characterised by the carrier
- A61K49/10—Organic compounds
- A61K49/14—Peptides, e.g. proteins
- A61K49/16—Antibodies; Immunoglobulins; Fragments thereof
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P1/00—Drugs for disorders of the alimentary tract or the digestive system
- A61P1/16—Drugs for disorders of the alimentary tract or the digestive system for liver or gallbladder disorders, e.g. hepatoprotective agents, cholagogues, litholytics
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P1/00—Drugs for disorders of the alimentary tract or the digestive system
- A61P1/18—Drugs for disorders of the alimentary tract or the digestive system for pancreatic disorders, e.g. pancreatic enzymes
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P3/00—Drugs for disorders of the metabolism
- A61P3/04—Anorexiants; Antiobesity agents
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P3/00—Drugs for disorders of the metabolism
- A61P3/06—Antihyperlipidemics
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P3/00—Drugs for disorders of the metabolism
- A61P3/08—Drugs for disorders of the metabolism for glucose homeostasis
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P3/00—Drugs for disorders of the metabolism
- A61P3/08—Drugs for disorders of the metabolism for glucose homeostasis
- A61P3/10—Drugs for disorders of the metabolism for glucose homeostasis for hyperglycaemia, e.g. antidiabetics
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P43/00—Drugs for specific purposes, not provided for in groups A61P1/00-A61P41/00
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P9/00—Drugs for disorders of the cardiovascular system
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P9/00—Drugs for disorders of the cardiovascular system
- A61P9/04—Inotropic agents, i.e. stimulants of cardiac contraction; Drugs for heart failure
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P9/00—Drugs for disorders of the cardiovascular system
- A61P9/10—Drugs for disorders of the cardiovascular system for treating ischaemic or atherosclerotic diseases, e.g. antianginal drugs, coronary vasodilators, drugs for myocardial infarction, retinopathy, cerebrovascula insufficiency, renal arteriosclerosis
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/475—Growth factors; Growth regulators
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/22—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against growth factors ; against growth regulators
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/24—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against cytokines, lymphokines or interferons
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K19/00—Hybrid peptides, i.e. peptides covalently bound to nucleic acids, or non-covalently bound protein-protein complexes
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/505—Medicinal preparations containing antigens or antibodies comprising antibodies
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/20—Immunoglobulins specific features characterized by taxonomic origin
- C07K2317/21—Immunoglobulins specific features characterized by taxonomic origin from primates, e.g. man
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/30—Immunoglobulins specific features characterized by aspects of specificity or valency
- C07K2317/33—Crossreactivity, e.g. for species or epitope, or lack of said crossreactivity
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/52—Constant or Fc region; Isotype
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/55—Fab or Fab'
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/56—Immunoglobulins specific features characterized by immunoglobulin fragments variable (Fv) region, i.e. VH and/or VL
- C07K2317/567—Framework region [FR]
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/76—Antagonist effect on antigen, e.g. neutralization or inhibition of binding
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/90—Immunoglobulins specific features characterized by (pharmaco)kinetic aspects or by stability of the immunoglobulin
- C07K2317/92—Affinity (KD), association rate (Ka), dissociation rate (Kd) or EC50 value
Landscapes
- Health & Medical Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Medicinal Chemistry (AREA)
- General Health & Medical Sciences (AREA)
- Organic Chemistry (AREA)
- Animal Behavior & Ethology (AREA)
- Public Health (AREA)
- Veterinary Medicine (AREA)
- Pharmacology & Pharmacy (AREA)
- Engineering & Computer Science (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Immunology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Nuclear Medicine, Radiotherapy & Molecular Imaging (AREA)
- Epidemiology (AREA)
- Genetics & Genomics (AREA)
- Biochemistry (AREA)
- Molecular Biology (AREA)
- Chemical Kinetics & Catalysis (AREA)
- General Chemical & Material Sciences (AREA)
- Biophysics (AREA)
- Diabetes (AREA)
- Endocrinology (AREA)
- Gastroenterology & Hepatology (AREA)
- Emergency Medicine (AREA)
- Obesity (AREA)
- Hematology (AREA)
- Zoology (AREA)
- Cardiology (AREA)
- Microbiology (AREA)
- Mycology (AREA)
- Heart & Thoracic Surgery (AREA)
- Toxicology (AREA)
- Radiology & Medical Imaging (AREA)
- Child & Adolescent Psychology (AREA)
- Urology & Nephrology (AREA)
- Biotechnology (AREA)
- Hospice & Palliative Care (AREA)
- Vascular Medicine (AREA)
Abstract
Изобретение относится к биохимии. Раскрыты антитело человека или антиген-связывающий фрагмент антитела человека, которые специфически связывают и ингибируют ангиопоэтин-подобный белок 4 человека (hANGPTL4). Описаны молекула нуклеиновой кислоты, кодидующая раскрытое антитело или антиген-связывающий фрагмент и вектор экспрессии, содержащий такую кислоту. Описан способ получения антитела, включающий использования клетки-хозяина, содержащий указанный вектор экспрессии. Также описана фармацевтическая композиция, содержащая раскрытое антитело. Представлен способ профилактики или лечения заболевания или нарушения, которое можно предотвратить, облегчить, сократить или ингибировать посредством сокращения или ингибирования активности ANGPTL4, включающий введение нуждающемуся в этом индивидууму терапевтически эффективного количества фармацевтической композиции. Изобретение можно использовать в лечении заболеваний или нарушений, связанных с ANGPTL4, таких как гиперлипидемия, гиперлипопротеинемия, дислипидемия, включая гипертриглицеридемию, гиперхолестеринемию, хиломикронемию и т.п. Далее анти-hANGPTL4 антитела можно вводить нуждающемуся в них индивидууму для профилактики или лечения заболеваний или нарушений, для которых нарушение метаболизма липидов представляет собой фактор риска. Такие заболевания или нарушения включают сердечно-сосудистые заболевания, такие как атеросклероз или атеросклеротическая болезнь сердца; острый панкреатит; неалкогольный стеатогепатит (NASH); диабет; ожирение; и т.п. 8 н. и 11 з.п. ф-лы, 7 ил., 18 табл., 13 пр.
Description
2420-187440RU/061
АНТИТЕЛА ЧЕЛОВЕКА ПРОТИВ АНГИОПОЭТИН-ПОДОБНОГО БЕЛКА 4 ЧЕЛОВЕКА
Область изобретения
Настоящее изобретение относится к антителам человека и антигенсвязывающим фрагментам антител человека, которые специфично связывают ангиопоэтин-подобный белок 4 человека (hANGPTL4), и к терапевтическим способам применения этих антител.
Связанные области
Липопротеинлипаза (LPL) играет центральную роль в метаболизме липопротеинов, поддерживая нормальные уровни липопротеинов в крови и, посредством тканеспецифической регуляции их активности, определяя, когда и в каких тканях высвобождаются триглицериды (TG). Опубликовано, что ANGPTL4 ингибирует LPL и замедляет катаболизм липопротеинов у людей и грызунов. Нулевые по ANGPTL4 мыши демонстрируют значительное снижение сывороточного уровня TG. И наоборот, инъекция мышам ANGPTL4 вызывает быстрое повышение уровней циркулирующих липидов, более интенсивное, чем при инъекции ангиопоэтин-подобного белка 3 (ANGPTL3) (Yoshida et al., 2002, J Lipid Res 43:1770-1772). Показано, что для ингибирования активности LPL и, таким образом, для индикации гипертриглицеридемии важна N-концевая суперспиральная область, а не C-концевой фибриноген-подобный домен белка ANGPTL4. Эти наблюдения показывают, что ингибирование ANGPTL4 может оказаться эффективным для лечения заболеваний, которые характеризуются повышенными уровнями липидов, включая первичную дислипидемию и гипертриглицеридемию, связанную с ожирением, метаболический синдром, диабет II типа и т.п. ANGPTL4 также играет роль в ангиогенезе и образовании злокачественных опухолей (Galaup et al., 2006, PNAS 103(49):18721-18726; Kim et al., 2000, Biochem J 346:603-610; и Ito et al., 2003, Cancer Res 63(20):6651-6657).
Последовательности нуклеиновых кислот и аминокислотные последовательности ANGPTL4 человека представлены в SEQ ID NO:475 и 476, соответственно. Антитела против ANGPTL4 описаны, например, в WO 2006/074228 и WO 2007/109307.
КРАТКАЯ СУЩНОСТЬ ИЗОБРЕТЕНИЯ
В первом аспекте изобретение относится к полностью моноклональным антителам (mAb) человека и их антигенсвязывающим фрагментам, которые специфически связывают ANGPTL4 (hANGPTL4) человека и нейтрализуют его активность.
Антитела (Ab) могут быть полноразмерными (например, антитела IgG1 или IgG4) или могут содержать только антигенсвязывающий участок (например, фрагмент Fab, F(ab')2 или scFv), и их можно модифицировать с изменением функциональности, например, для устранения конечных эффекторных функций (Reddy et al., 2000, J. Immunol. 164:1925-1933).
В одном из вариантов осуществления изобретение включает антитело или антигенсвязывающий фрагмент антитела, содержащие вариабельную область тяжелой цепи (HCVR), выбранную из группы, состоящей из SEQ ID NO: 2, 18, 22, 26, 42, 46, 50, 66, 70, 74, 90, 94, 98, 114, 118, 122, 138, 142, 146, 162, 166, 170, 186, 190, 194, 210, 214, 218, 234, 238, 242, 258, 262, 266, 282, 286, 290, 306, 310, 314, 330, 334, 338, 354, 358, 362, 378, 382, 386, 402, 406, 410, 426, 430, 434, 450, 454, 458, 466, 468 и 487 или в значительной степени сходных последовательностей, идентичных по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или, по меньшей мере на 99%. В другом варианте осуществления антитело или его антигенсвязывающий фрагмент содержат HCVR с аминокислотной последовательностью, выбранной из группы, состоящей из SEQ ID NO:26, 42, 46, 487, 74, 90, 94, 122, 138, 142, 146, 162 и 166. В еще одном варианте осуществления антитело или его фрагмент содержат HCVR, содержащую SEQ ID NO:42, 487, 90, 138 или 162.
В одном из вариантов осуществления антитело или антигенсвязывающий фрагмент антитела содержат вариабельную область легкой цепи (LCVR), выбранную из группы, состоящей из SEQ ID NO: 10, 20, 24, 34, 44, 48, 58, 68, 72, 82, 92, 96, 106, 116, 120, 130, 140, 144, 154, 164, 168, 178, 188, 192, 202, 212, 216, 226, 236, 240, 250, 260, 264, 274, 284, 288, 298, 308, 312, 322, 332, 336, 346, 356, 360, 370, 380, 384, 394, 404, 408, 418, 428, 432, 442, 452 и 456 или в значительной степени сходных последовательностей, идентичных по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или, по меньшей мере на 99%. В другом варианте осуществления антитело или антигенсвязывающий участок антитела содержат LCVR с аминокислотной последовательностью, выбранной из группы, состоящей из SEQ ID NO:34, 44, 48, 82, 92, 96, 130, 140, 144, 154, 164 и 168. В еще одном варианте осуществления антитело или его фрагмент содержат LCVR, содержащую SEQ ID NO:44, 92, 140 или 164.
В дополнительных вариантах осуществления антитело или его фрагмент содержат пару последовательностей HCVR и LCVR (HCVR/LCVR), выбранную из группы, состоящей из SEQ ID NO:2/10, 18/20, 22/24, 26/34, 42/44, 487/44, 46/48, 50/58, 66/68, 70/72, 74/82, 90/92, 94/96, 98/106, 114/116, 118/120, 122/130, 138/140, 142/144, 146/154, 162/164, 166/168, 170/178, 186/188, 190/192, 194/202, 210/212, 214/216, 218/226, 234/236, 238/240, 242/250, 258/260, 262/264, 266/274, 282/284, 286/288, 290/298, 306/308, 310/312, 314/322, 330/332, 334/336, 338/346, 354/356, 358/360, 362/370, 378/380, 382/384, 386/394, 402/404, 406/408, 410/418, 426/428, 430/432, 434/442, 450/452, 454/456, 458/394, 466/404 и 468/408. В одном из вариантов осуществления антитело или его фрагмент содержат HCVR и LCVR, выбранные из пар аминокислотных последовательностей SEQ ID NO:26/34, 42/44, 487/44, 46/48, 74/82, 90/92, 94/96, 122/130, 138/140, 142/144, 146/154, 162/164 и 166/168. В другом варианте осуществления антитело или его фрагмент содержат пару HCVR/LCVR, содержащую SEQ ID NO:42/44, 487/44, 90/92, 138/140 или 162/164.
Во втором аспекте изобретение предоставляет антитело или антигенсвязывающий фрагмент антитела, содержащие аминокислотную последовательность определяющей комплементарность области тяжелой цепи 3 (HCDR3), выбранную из группы, состоящей из SEQ ID NO: 8, 32, 56, 80, 104, 128, 152, 176, 200, 224, 248, 272, 296, 320, 344, 368, 392, 416, 440 и 464 или в значительной степени сходных последовательностей, идентичных по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или, по меньшей мере на 99%; и аминокислотную последовательность легкой цепи CDR3 (LCDR3), выбранную из группы, состоящей из SEQ ID NO:16, 40, 64, 88, 112, 136, 160, 184, 208, 232, 256, 280, 304, 328, 352, 376, 400, 424 и 448 или в значительной степени сходных последовательностей, идентичных по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или, по меньшей мере на 99%. В одном из вариантов осуществления антитело или его фрагмент содержат пару аминокислотных последовательностей HCDR3/LCDR3, содержащую SEQ ID NO:32/40, 80/88, 128/136 или 152/160. В другом варианте осуществления антитело или его фрагмент содержат пару аминокислотных последовательностей HCDR3/LCDR3, содержащую SEQ ID NO:32/40 или 80/88.
В дополнительном варианте осуществления антитело или его фрагмент дополнительно содержат аминокислотную последовательность тяжелой цепи CDR1 (HCDR1), выбранную из группы, состоящей из SEQ ID NO:4, 28, 52, 76, 100, 124, 148, 172, 196, 220, 244, 268, 292, 316, 340, 364, 388, 412, 436 и 460 или в значительной степени сходных последовательностей, идентичных по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99%; и аминокислотную последовательность тяжелой цепи CDR2 (HCDR2), выбранную из группы, состоящей из SEQ ID NO:6, 30, 54, 78, 102, 126, 150, 174, 198, 222, 246, 270, 294, 318, 342, 366, 390, 414, 438 и 462 или в значительной степени сходных последовательностей, идентичных по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или, по меньшей мере на 99%; и необязательно дополнительно содержат аминокислотную последовательность легкой цепи CDR1 (LCDR1), выбранную из группы, состоящей из SEQ ID NO:12, 36, 60, 84, 108, 132, 156, 180, 204, 228, 252, 276, 300, 324, 348, 372, 396, 420 и 444 или в значительной степени сходных последовательностей, идентичных по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99%; и/или аминокислотную последовательность легкой цепи CDR2 (LCDR2), выбранную из группы, состоящей из SEQ ID NO:14, 38, 62, 86, 1 10, 134, 158, 182, 206, 230, 254, 278, 302, 326, 350, 374, 398, 422 и 446 или в значительной степени сходных последовательностей, идентичных по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99%.
Альтернативно, изобретение предоставляет антитело или антигенсвязывающий фрагмент антитела, содержащие комбинацию HCDR1/HCDR2/HCDR3, выбранную из группы, состоящей из SEQ ID NO: 4/6/8, 28/30/32, 52/54/56, 76/78/80, 100/102/104, 124/126/128, 148/150/152, 172/174/176, 196/198/200, 220/222/224, 244/246/248, 268/270/272, 292/294/296, 316/318/320, 340/342/344, 364/366/368, 388/390/392, 412/414/416, 436/438/440 и 460/462/464; и/или комбинацию LCDR1/LCDR2/LCDR3, выбранную из группы, состоящей из SEQ ID NO:12/14/16, 36/38/40, 60/62/64, 84/86/88, 108/110/112, 132/134/136, 156/158/160, 180/182/184, 204/206/208, 228/230/232, 252/254/256, 276/278/280, 300/302/304, 324/326/328, 348/350/352, 372/374/376, 396/398/400, 420/422/424 и 444/446/448.
В одном из вариантов осуществления HCDR1, HCDR2 и HCDR3 выбраны из группы, состоящей из SEQ ID NO:28/30/32, 76/78/80, 124/126/128, и 148/150/152; и/или LCDR1, LCDR2 и LCDR3 выбраны из группы, состоящей из SEQ ID NO:36/38/40, 84/86/88, 132/134/136 и 156/158/160. В еще одном варианте осуществления аминокислотные последовательности тяжелой и легкой цепи CDR содержат комбинацию последовательностей CDR, выбранную из группы, состоящей из SEQ ID NO:28/30/32/36/38/40, 76/78/80/84/86/88, 124/126/128/132/134/136 и 148/150/152/156/158/160. В еще одном варианте осуществления антитело или его антигенсвязывающий фрагмент содержат последовательности CDR тяжелой и легкой цепи SEQ ID NO: 28/30/32/36/38/40 или 76/78/80/84/86/88.
В связанном варианте осуществления изобретение содержит антитело или антигенсвязывающий фрагмент антитела, которые специфически связываются с hANGPTL4, где антитело или его фрагмент содержат домены CDR тяжелой и легкой цепи, содержащиеся в паре HCVR/LCVR, выбранной из группы, состоящей из SEQ ID NO:2/10, 18/20, 22/24, 26/34, 42/44, 487/44, 46/48, 50/58, 66/68, 70/72, 74/82, 90/92, 94/96, 98/106, 114/116, 118/120, 122/130, 138/140, 142/144, 146/154, 162/164, 166/168, 170/178, 186/188, 190/192, 194/202, 210/212, 214/216, 218/226, 234/236, 238/240, 242/250, 258/260, 262/264, 266/274, 282/284, 286/288, 290/298, 306/308, 310/312, 314/322, 330/332, 334/336, 338/346, 354/356, 358/360, 362/370, 378/380, 382/384, 386/394, 402/404, 406/408, 410/418, 426/428, 430/432, 434/442, 450/452, 454/456, 458/394, 466/404 и 468/408. Способы и техника обнаружения CDR в аминокислотных последовательностях HCVR и LCVR известны в данной области, и их можно применять для обнаружения CDR в определенных аминокислотных последовательностях HCVR и/или LCVR, описываемых в настоящем документе. Общепринятые определения, которые можно применять для определения границ CDR, включают определение по Kabat, определение по Chothia и определение AbM. Вкратце, определение по Kabat основано на вариабельности последовательности, определение по Chothia основано на определении местоположения структурных петлевых участков, а определение AbM совмещает подходы по Kabat и по Chothia. См., например, Kabat, "Sequences of Proteins of Immunological Interest", National Institutes of Health, Bethesda, Md. (1991); Al-Lazikani et al., J. Mol. Biol. 273:927-948 (1997); и Martin et al., Proc. Natl. Acad. Sci. USA 86:9268-9272 (1989). Также для обнаружения последовательностей CDR в антителе можно использовать общедоступные базы данных. В одном из вариантов осуществления антитело или его фрагмент содержат последовательности CDR, содержащиеся в паре HCVR и LCVR, выбранной из группы, состоящей из пар аминокислотных последовательностей SEQ ID NO:26/34, 42/44, 487/44, 46/48, 74/82, 90/92, 94/96, 122/130, 138/140, 142/144, 146/154, 162/164 и 166/168. В другом варианте осуществления антитело или его фрагмент содержат последовательности CDR, содержащиеся в паре последовательностей HCVR и LCVR SEQ ID NO:42/44, 487/44, 90/92, 138/140 или 162/164.
В другом связанном варианте осуществления изобретение относится к антителу или его антигенсвязывающему фрагменту, которые конкурируют за специфическое связывание с hANGPTL4 с антителом или антигенсвязывающим фрагментом, содержащим последовательности CDR тяжелой и легкой цепи SEQ ID NO:28/30/32/36/38/40, 76/78/80/84/86/88, 124/126/128/132/134/136, или 148/150/152/156/158/160. В одном из вариантов осуществления антитело или антигенсвязывающий фрагмент по изобретению конкурирует за специфическое связывание с hANGPTL4 с антителом, содержащим пару последовательностей HCVR/LCVR SEQ ID NO:42/44, 487/44, 90/92, 138/140 или 162/164.
В другом связанном варианте осуществления изобретение относится к антителу или его антигенсвязывающему фрагменту, связывающим тот же эпитоп на hANGPTL4, который распознается антителом или его фрагментом, содержащим последовательности CDR тяжелой и легкой цепи SEQ ID NO:28/30/32/36/38/40, 76/78/80/84/86/88, 124/126/128/132/134/136 или 148/150/152/156/158/160. В одном из вариантов осуществления антитело или антигенсвязывающий фрагмент по изобретению распознают эпитоп на hANGPTL4, который распознается антителом, содержащим пару последовательностей HCVR/LCVR SEQ ID NO:42/44, 487/44, 90/92, 138/140 или 162/164.
В третьем аспекте изобретение относится к молекулам нуклеиновой кислоты, кодирующим антитела против ANGPTL4 или их фрагменты, в частности, описанные выше. Рекомбинантные векторы экспрессии, несущие нуклеиновые кислоты по изобретению, и клетки-хозяева, например, бактериальные клетки, такие как E. coli, или клетки млекопитающих, такие как клетки CHO, в которые встраивают такие векторы, также включены в изобретение, наряду со способами получения антител посредством культивирования клеток-хозяев в условиях, обеспечивающих получение антител и извлечение полученных антител.
В одном из вариантов осуществления изобретение относится к антителу или его фрагменту, содержащим HCVR, кодируемую нуклеиновой кислотой с последовательностью, выбранной из группы, состоящей из SEQ ID NO:1, 17, 21, 25, 41, 45, 49, 65, 69, 73, 89, 93, 97, 113, 117, 121, 137, 141, 145, 161, 165, 169, 185, 189, 193, 209, 213, 217, 233, 237, 241, 257, 261, 265, 281, 285, 289, 305, 309, 313, 329, 333, 337, 353, 357, 361, 377, 381, 385, 401, 405, 409, 425, 429, 433, 449, 453, 457, 465, 467 и 486 или в значительной степени сходных последовательностей, гомологичных по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99%. В другом варианте осуществления антитело или его фрагмент содержат HCVR, кодируемую нуклеиновой кислотой с последовательностью, выбранной из группы, состоящей из SEQ ID NO:25, 41, 45, 73, 89, 93, 121, 137, 141, 145, 161, 165 и 486. В еще одном варианте осуществления антитело или его фрагмент содержат HCVR, кодируемую нуклеиновой кислотой с последовательностью SEQ ID NO: 41, 89, 137, 161 или 486.
В одном из вариантов осуществления антитело или его антигенсвязывающий фрагмент содержат LCVR, кодируемую нуклеиновой кислотой с последовательностью, выбранной из группы, состоящей из SEQ ID NO:9, 19, 23, 33, 43, 47, 57, 67, 71, 81, 91, 95, 105, 115, 119, 129, 139, 143, 153, 163, 167, 177, 187, 191, 201, 211, 215, 225, 235, 239, 249, 259, 263, 273, 283, 287, 297, 307, 311, 321, 331, 335, 345, 355, 359, 369, 379, 383, 393, 403, 407, 417, 427, 431, 441, 451 и 455 или в значительной степени сходных последовательностей, гомологичных по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99%. В другом варианте осуществления антитело или его фрагмент содержат LCVR, кодируемую нуклеиновой кислотой с последовательностью, выбранной из группы, состоящей из SEQ ID NO:33, 43, 47, 81, 91, 95, 129, 139, 143, 153, 163 и 167. В еще одном варианте осуществления антитело или его фрагмент содержат LCVR, кодируемую нуклеиновой кислотой с последовательностью SEQ ID NO: 43, 91, 139 или 163.
В дополнительных вариантах осуществления антитело или его фрагмент содержат пару последовательностей HCVR и LCVR (HCVR/LCVR), кодируемую парой нуклеиновых кислот с последовательностями, выбранными из группы, состоящей из SEQ ID NO: 1/9, 17/19, 21/23, 25/33, 41/43, 486/43, 45/47, 49/57, 65/67, 69/71, 73/81, 89/91, 93/95, 97/105, 113/115, 117/119, 121/129, 137/139, 141/143, 145/153, 161/163, 165/167, 169/177, 185/187, 189/191, 193/201, 209/211, 213/215, 217/225, 233/235, 237/239, 241/249, 257/259, 261/263, 265/273, 281/283, 285/287, 289/297, 305/307, 309/311, 313/321, 329/331, 333/335, 337/345, 353/355, 357/359, 361/369, 377/379, 381/383, 385/393, 401/403, 405/407, 409/417, 425/427, 429/431, 433/441, 449/451, 453/455, 457/393, 465/403 и 467/407. В одном из вариантов осуществления антитело или его фрагмент содержат пару последовательностей HCVR/LCVR, кодируемую парой нуклеиновых кислот с последовательностями, выбранными из группы, состоящей из SEQ ID NO:25/33, 41/43, 486/43, 45/47, 73/81, 89/91, 93/95, 121/129, 137/139, 141/143, 145/153, 161/163 и 165/167. В еще одном варианте осуществления антитело или его фрагмент содержат пару HCVR/LCVR, кодируемую парой нуклеиновых кислот с последовательностями SEQ ID NO:41/43, 486/43, 89/91, 137/139 или 161/163.
В одном из вариантов осуществления изобретение относится к антителу или антигенсвязывающему фрагменту антитела, содержащим домен HCDR3, кодируемый нуклеотидной последовательностью, выбранной из группы, состоящей из SEQ ID NO:7, 31, 55, 79, 103, 127, 151, 175, 199, 223, 247, 271, 295, 319, 343, 367, 391, 415, 439 и 463 или в значительной степени сходными последовательностями, гомологичными по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99%; и домен LCDR3, кодируемый нуклеотидной последовательностью, выбранной из группы, состоящей из SEQ ID NO:15, 39, 63, 87, 111, 135, 159, 183, 207, 231, 255, 279, 303, 327, 351, 375, 399, 423 и 447 или в значительной степени сходными последовательностями, гомологичными по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99%. В другом варианте осуществления антитело или его фрагмент содержат пару последовательностей HCDR3 и LCDR3, кодируемых парой нуклеиновых кислот с последовательностями SEQ ID NO:31/39, 79/87, 127/135 или 151/159. В еще одном варианте осуществления антитело или его фрагмент содержат пару последовательностей HCDR3 и LCDR3, кодируемых парой нуклеиновых кислот с последовательностями SEQ ID NO:31/39 или 79/87.
В дополнительном варианте осуществления антитело или его фрагмент дополнительно содержат домен HCDR1, кодируемый нуклеотидной последовательностью, выбранной из группы, состоящей из SEQ ID NO:3, 27, 51, 75, 99, 123, 147, 171, 195, 219, 243, 267, 291, 315, 339, 363, 387, 411, 435 и 459 или в значительной степени сходными последовательностями, гомологичными по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99%; и домен HCDR2, кодируемый нуклеотидной последовательностью, выбранной из группы, состоящей из SEQ ID NO:5, 29, 53, 77, 101, 125, 149, 173, 197, 221, 245, 269, 293, 317, 341, 365, 389, 413, 437 и 461 или в значительной степени сходными последовательностями, гомологичными по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99%; и необязательно дополнительно содержит домен LCDR1, кодируемый нуклеотидной последовательностью, выбранной из группы, состоящей из SEQ ID NO:11, 35, 59, 83, 107, 131, 155, 179, 203, 227, 251, 275, 299, 323, 347, 371, 395, 419 и 443 или в значительной степени сходными последовательностями, гомологичными по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99%; и/или домен LCDR2, кодируемый нуклеотидной последовательностью, выбранной из группы, состоящей из SEQ ID NO:13, 37, 61, 85, 109, 133, 157, 181, 205, 229, 253, 277, 301, 325, 349, 373, 397, 421 и 445 или в значительной степени сходными последовательностями, гомологичными, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99%.
Альтернативно, изобретение относится к антителу или антигенсвязывающему фрагменту антитела, содержащим комбинацию HCDR1/HCDR2/HCDR3, кодируемую комбинацией нуклеотидных последовательностей, выбранной из группы, состоящей из SEQ ID NO:3/5/7, 27/29/31, 51/53/55, 75/77/79, 99/101/103, 123/125/127, 147/149/151, 171/173/175, 195/197/199, 219/221/223, 243/245/247, 267/269/271, 291/293/295, 315/317/319, 339/341/343, 363/365/367, 387/389/391, 411/413/415, 435/437/439 и 459/461/463; и/или комбинацию LCDR1/LCDR2/LCDR3, кодируемую комбинацией нуклеотидных последовательностей, выбранной из группы, состоящей из SEQ ID NO:11/13/15, 35/37/39, 59/61/63, 83/85/87, 107/109/111, 131/133/135, 155/157/159, 179/181/183, 203/205/207, 227/229/231, 251/253/255, 275/277/279, 299/301/303, 323/325/327, 347/349/351, 371/373/375, 395/397/399, 419/421/423 и 443/445/447.
В одном из вариантов осуществления HCDR1, HCDR2 и HCDR3 кодируются комбинацией нуклеотидных последовательностей, выбранной из группы, состоящей из SEQ ID NO:27/29/31, 75/77/79, 123/125/127 и 147/149/151; и/или LCDR1, LCDR2 и LCDR3 кодируются комбинацией нуклеотидных последовательностей, выбранной из группы, состоящей из SEQ ID NO:35/37/39, 83/85/87, 131/133/135, и 155/157/159. В еще одном варианте осуществления антитело или его фрагмент содержат последовательности CDR тяжелой и легкой цепи, кодируемые комбинацией нуклеотидных последовательностей, выбранной из группы, состоящей из SEQ ID NO: 27/29/31/35/37/39; 75/77/79/83/85/87; 123/125/127/131/133/135; и 147/149/151/155/157/159. В другом варианте осуществления антитело или его антигенсвязывающий участок содержат последовательности CDR тяжелой и легкой цепи, кодируемые комбинацией нуклеотидных последовательностей SEQ ID NO: 27/29/31/35/37/39; или 75/77/79/83/85/87.
Четвертый аспект изобретение относится к выделенному антителу или антигенсвязывающему фрагменту антитела, которые специфически связываются с hANGPTL4 и содержат HCDR3 и LCDR3, где HCDR3 содержит аминокислотную последовательность формулы X1-X2-X3-X4-X5-X6-X7-X8-X9-X10-X11-X12-X13-X14-X15-X16-X17-X18-X19-X20 (SEQ ID NO:471), где X1 представляет собой Ala, X2 представляет собой Arg или Lys, X3 представляет собой Gly или Glu, X4 представляет собой Gly, Asp или пропуск, X5 представляет собой Asp или пропуск, X6 представляет собой Leu, Arg или пропуск, X7 представляет собой Arg или Ser, X8 представляет собой Phe, Gly или Arg, X9 представляет собой Leu, His или Asn, X10 представляет собой Asp, Pro или Tyr, X11 представляет собой Trp, Tyr или Phe, X12 представляет собой Leu, Phe, Val или Asp, X13 представляет собой Ser, Tyr или Gly, X14 представляет собой Ser, Tyr или Asp, X15 представляет собой Tyr, X16 представляет собой Phe или Gly, X17 представляет собой Leu или пропуск, X18 представляет собой Asp, X19 представляет собой Tyr, Val или Phe и X20 представляет собой Trp; и LCDR3 содержит аминокислотную последовательность формулы X1-X2-X3-X4-X5-X6-X7-X8-X9-X10 (SEQ ID NO:474), где X1 представляет собой Gln, X2 представляет собой Asn или Gln, X3 представляет собой Tyr или Leu, X4 представляет собой Asn, His, Ser или Asp, X5 представляет собой Thr или Ser, X6 представляет собой Ala или Tyr, X7 представляет собой Pro, Ser или Phe, X8 представляет собой Leu или Arg, X9 представляет собой Thr и X10 представляет собой Phe.
В дополнительном варианте осуществления антитело или его фрагмент дополнительно содержит последовательность HCDR1, содержащую аминокислотную последовательность формулы X1-X2-X3-X4-X5-X6-X7-X8 (SEQ ID NO:469), где X1 представляет собой Gly, X2 представляет собой Gly или Phe, X3 представляет собой Ser или Thr, X4 представляет собой Phe, X5 представляет собой Ser, X6 представляет собой Ile, Ser или Thr, X7 представляет собой His или Tyr и X8 представляет собой His, Gly или Asp; последовательность HCDR2, содержащую аминокислотную последовательность формулы X1-X2-X3-X4-X5-X6-X7-X8 (SEQ ID NO:470), где X1 представляет собой Ile, X2 представляет собой Asn, Ser или Gly, X3 представляет собой His, Phe, Ser или Val, X4 представляет собой Arg, Asp или Ala, X5 представляет собой Gly, X6 представляет собой Gly или пропуск, X7 представляет собой Ser, Asn или Asp и X8 представляет собой Thr или Lys; последовательность LCDR1, содержащую аминокислотную последовательность формулы X1-X2-X3-X4-X5-X6 (SEQ ID NO:472), где X1 представляет собой Gln, X2 представляет собой Gly или Ser, X3 представляет собой Ile, X4 представляет собой Ser или Asn, X5 представляет собой Asp, Ser или Arg и X6 представляет собой Tyr или Trp; и последовательность LCDR2, содержащую аминокислотную последовательность формулы X1-X2-X3 (SEQ ID NO:473), где X1 представляет собой Ala или Lys, X2 представляет собой Ala и X3 представляет собой Ser. Выравнивания последовательностей моноклональных антител H1H268P, H1H284P, H1H291P и H1H292P показаны на фиг. 1 (HCVR) и фиг. 2 (LCVR).
В пятом аспекте изобретение относится к антителу человека против ANGPTL4 или его антигенсвязывающему фрагменту, содержащим вариабельную область тяжелой цепи (HCVR), кодируемую сегментами нуклеотидной последовательности, полученными из последовательностей зародышевых линий VH, DH и JH, и вариабельную область легкой цепи (LCVR), кодируемую сегментами нуклеотидной последовательности, полученными из последовательностей зародышевых линий VK и JK, где HCVR и LCVR кодируются сегментами нуклеотидной последовательности, полученными из комбинаций генов зародышевых линий, выбранных из группы, состоящей из: (i) VH3-30, DH5-12, JH6, VK1-9 и JK4; (ii) VH4-34, DH3-3, JH4, VK1-27 и JK4; и (iii) VH3-13, DH1-26, JH4, VK1-5 и JK1.
В шестом аспекте изобретение относится к антителу или его антигенсвязывающему фрагменту, которые специфически связываются с hANGPTL4 с константой диссоциации (KD), составляющей приблизительно 1 нМ или менее, как показано посредством поверхностного плазмонного резонанса (например, BIACORE™). В определенных вариантах осуществления антитело по изобретению характеризуется KD, составляющей приблизительно 500 пМ или менее; приблизительно 400 пМ или менее; приблизительно 300 пМ или менее; приблизительно 200 пМ или менее; приблизительно 150 пМ или менее; приблизительно 100 пМ или менее; или приблизительно 50 пМ или менее.
В седьмом аспекте настоящее изобретение относится к антителу против hANGPTL4 или его антигенсвязывающему фрагменту, которые связывают белок hANGPTL4 SEQ ID NO:476, но не проявляют перекрестной реактивности в отношении родственного белка, такого как ангиопоэтин-подобный белок 3 человека (hANGPTL3; SEQ ID NO:485), как показано, например, посредством ELISA, поверхностного плазмонного резонанса или способа LUMINEX® XMAP®, как описано в настоящем документе. ANGPTL3 представляет собой другой секретируемый белок, который, как показано, сокращает активность LPL и содержит N-концевой суперспиральный участок и C-концевой фибриноген-подобный домен (Ono et al., 2003, J Biol Chem 43:41804-41809). В связанных вариантах осуществления изобретение относится к антителу против hANGPTL4 или его антигенсвязывающему фрагменту, которые связывают белок hANGPTL4 и проявляют перекрестную реактивность в отношении белка hANGPTL3. В определенных вариантах осуществления аффинность связывания антитела hANGPTL4 или его фрагмента с белком hANGPTL3 составляет приблизительно 75% или менее, или приблизительно 50% или менее от аффинности связывания антитела или его фрагмента с белком hANGPTL4. В другом связанном варианте осуществления изобретение относится к антителу против hANGPTL4 или его антигенсвязывающему фрагменту, которые не проявляют перекрестной реактивности в отношении ANGPTL4 мыши (mANGPTL4: SEQ ID NO:478), но проявляют перекрестную реактивность в отношении ANGPTL4 яванского макака (Macaca fascicularis; аминокислотная последовательность N-концевых 1-148 остатков и последовательности кодирующих ДНК показаны в SEQ ID NO:490 и 489, соответственно) и/или ANGPTL4 макака-резуса (Macaca mulatta; аминокислотная последовательность N-концевых 1-148 остатков и последовательности кодирующих ДНК показаны в SEQ ID NO:492 и 491, соответственно).
Настоящее изобретение относится к антителам против hANGPTL4, характеризующимся модифицированным паттерном гликозилирования. В некоторых областях применения модификацию можно применять для удаления нежелательных участков гликозилирования, или например, для удаления составной группы фукозы для усиления антитело-зависимой клеточной цитотоксичности (ADCC) (см. Shield et al. (2002) JBC 277:26733). В других областях применения удаление N-участка гликозилирования может уменьшать нежелательный иммунный ответ на терапевтические антитела или повышать аффинность антител. В других областях применения можно применять модификацию галактозилирования для модифицирования обусловленной комплементом цитотоксичности (CDC).
В восьмом аспекте изобретение относится к фармацевтической композиции, содержащей рекомбинантное антитело человека или его фрагмент, которые специфически связываются с hANGPTL4, и фармацевтически приемлемый носитель. В одном из вариантов осуществления изобретение относится к композиции, которая представляет собой комбинацию антитела или его антигенсвязывающего фрагмента по изобретению и второго терапевтического средства. Второе терапевтическое средство может представлять собой одно или несколько из таких средств как (1) ингибиторы 3-гидрокси-3-метилглутарил-кофермент A (HMG-CoA) редуктазы, такие как церивастатин, аторвастатин, симвастатин, питавастатин, розувастатин, флувастатин, ловастатин, правастатин и т.п.; (2) ингибиторы захвата холестерина и/или повторного всасывания желчной кислоты; (3) ниацин, который усиливает катаболизм липопротеинов; (4) фибраты или амфипатические карбоновые кислоты, которые снижают уровень липопротеинов низкой плотности (LDL), увеличивают уровень липопротеинов высокой плотности (HDL) и TG и снижают частоту возникновения несмертельного инфаркта миокарда; и (5) активаторы фактора транскрипции LXR, участвующего в разрушении холестерина, такие как 22-гидроксихолестерол, или фиксированные комбинации, такие как эзетимиб плюс симвастатин; статин с секвестрантами желчных кислот (например, холестирамин, колестипол, колесевелам), фиксированная комбинация ниацин плюс статин (например, ниацин с ловастатином); или с другими гиполипидемическими средствами, такими как сложные эфиры омега-3-жирных кислот (например, омакор). Кроме того, второе терапевтическое средство может представлять собой один или несколько других ингибиторов ANGPTL4, а также ингибиторы других молекул, таких как ANGPTL3, ANGPTL5, ANGPTL6 и пропротеинконвертаза субтилизин/кексин типа 9 (PCSK9), которые вовлечены в метаболизм липидов, в частности, в гомеостаз холестерина и/или триглицеридов. Ингибиторы этих молекул включают низкомолекулярные соединения и антитела, которые специфически связываются с этими молекулами и блокируют их активность.
В связанных вариантах осуществления второе терапевтическое средство может представлять собой одно или несколько противоопухолевых средств, таких как химиотерапевтические средства, антиангиогенные средства, ингибирующие рост средства, цитотоксические средства, апоптозные средства и другие средства, хорошо известные в данной области как средства для лечения злокачественной опухоли или других пролиферативных заболеваний или нарушений, а также другие терапевтические средства, такие как анальгетики, противовоспалительные средства, включая нестероидные противовоспалительные лекарственные средства (NSAIDS), такие как ингибиторы Cox-2 и т.п., позволяющие облегчить и/или сократить симптомы, сопутствующие злокачественной опухоли.
В девятом аспекте изобретение предоставляет способы ингибирования активности hANGPTL4 с применением антитела к hANGPTL4 или антигенсвязывающего участка антитела по изобретению, где терапевтические способы включают введение терапевтически эффективного количества фармацевтической композиции, содержащей антитело или антигенсвязывающий фрагмент антитела по изобретению и необязательно одно или несколько дополнительных терапевтических средств, описанных выше. Заболевание или нарушение, подлежащее лечению, представляет собой любое заболевание или состояние, которое можно облегчить, ослабить, ингибировать или предотвратить или сократить частоту возникновения по сравнению с отсутствием лечения с применением антитела к hANGPTL4 (например, ANGPTL4-обусловленные заболевания или нарушения), посредством ингибирования или снижения активности ANGPTL4. Примеры заболеваний или нарушений, которые можно лечить способами по изобретению, в качестве неограничивающих примеров включают заболевания и нарушения, затрагивающие метаболизм липидов, такие как гиперлипидемия, гиперлипопротеинемия и дислипидемия, включая атерогенную дислипидемию, диабетическую дислипидемию, гипертриглицеридемию, включая тяжелую гипертриглицеридемию с TG > 1000 мг/дл, гиперхолестеринемию, хиломикронемию, смешанную дислипидемию (ожирение, метаболический синдром, диабет и т.д.), липодистрофию, липоатрофию и т.п., которые вызываются, например, сниженной активностью LPL и/или дефицитом LPL, сниженной активностью рецепторов LDL и/или дефицитом рецепторов LDL, нарушениями ApoC2, дефицитом ApoE, повышенным ApoB, повышенным выделением и/или сниженным разрушением липопротеинов с очень низкой плотностью (VLDL), воздействием определенного лекарственного средства (например, дислипидемия, вызванная лечением глюкокортикоидами), любой генетической предрасположенностью, питанием, образом жизни и т.п. Способы по изобретению также могут предотвращать или лечить заболевания или нарушения, связанные с или вызванные гиперлипидемией, гиперлипопротеинемией и/или дислипидемией, включая в качестве неограничивающих примеров сердечно-сосудистые заболевания или нарушения, такие как атеросклероз, аневризмы, гипертензия, ангина, инсульт, цереброваскулярная болезнь, застойная сердечная недостаточность, ишемическая болезнь сердца, инфаркт миокарда, заболевания периферических сосудов и т.п.; острый панкреатит; неалкогольный стеатогепатит (NASH); нарушения, связанные с содержанием сахара в крови, такие как диабет; ожирение и т.п.
Другие примеры заболеваний или нарушений, которые можно лечить способами по изобретению, включают злокачественную опухоль, а также не неопластические ангиогенез-связанные заболевания или нарушения, включая глазные ангиогенные заболевания или нарушения, такие как связанная с возрастом дегенерация желтого пятна, окклюзия центральной вены сетчатки или окклюзия ветки вены сетчатки, диабетическая ретинопатия, ретинопатия недоношенных и т.п., воспалительные заболевания или нарушения, такие как артрит, ревматоидный артрит (RA), псориаз и т.п.
Другие варианты осуществления предоставлены в приведенном ниже подробном описании.
КРАТКОЕ ОПИСАНИЕ ФИГУР
На фиг. 1 представлено выравнивание последовательностей вариабельных областей тяжелой цепи (HCVR) антител H1H268P, H1H284P, H1H291P и H1H292P.
На фиг. 2 представлено выравнивание последовательностей вариабельных областей легкой цепи (LCVR) антител H1H268P, H1H284P, H1H291P и H1H292P.
На фиг. 3A и 3B показан фармакокинетический клиренс антител против ANGPTL4 у мышей дикого типа (фиг. 3A) и у трансгенных мышей, экспрессирующих ANGPTL4 человека [мыши hAngptl4(+/+); или "гуманизированные ANGPTL4 мыши"] (фиг. 3B). H4H268P2 (□); H4H284P (▲); и контрольные hIgG4 (●).
На фиг. 4 показано воздействие антител против ANGPTL4, H4H268P2, на сывороточный уровень триглицеридов (TG) у гуманизированных ANGPTL4 мышей, скрещенных с нулевыми по ApoE мыши. Показан процент (%) изменений сывороточного уровня TG, вызванных H4H268P2, по сравнению с контрольным антителом с неподходящей специфичностью. Контрольные Ab (○); и H4H268P2 (■).
На фиг. 5 показано воздействие антитела против ANGPTL4 H4H268P2 и снижающего уровень TG лекарственного средства фенофибрат, как по отдельности, так и в комбинации, на сывороточный уровень TG у гуманизированных ANGPTL4 мышей.
На фиг. 6 представлены результаты этапа I пилотного исследования воздействия антител против ANGPTL4 на снижение сывороточного уровня TG, а также других липидов, у страдающих ожирением макак-резусов (Macaca mulatta). Все обезьяны получали носитель (10 мМ гистидин, pH 6) внутривенным вливанием на сутки -5 и либо H4H268P2 (n=3) (●), либо H4H284P (n=3) (□) в дозе 10 мг/кг внутривенно на сутки 0. Образцы сыворотки собирали, начиная с исходного периода по сутки 35 после лечения. Средние исходные уровни для каждого животного определяли на основе образцов, собранных на сутки -7, -5 и 0, и для каждой группы Ab определяли и усредняли процент (%) изменений сывороточных уровней TG по сравнению с исходными значениями.
На фиг. 7 представлены результаты этапа II пилотного исследования воздействия антитела против ANGPTL4 H4H268P2 на снижение сывороточного уровня TG у страдающих ожирением обезьян, как описано для фиг. 6, за исключением того, что этап введения носителя не проводили. Для каждой обезьяны получали средние исходные уровни на основе образцов, собранных на сутки -7, -3 и 0. Обезьян разделяли на группы на основе их исходных уровней: A. TG<150 мг/дл (n=3;□); B. 150 мг/дл<TG<500 мг/дл (n=4; ●); и C. TG>1000 мг/дл (n=1; ∇). Процент (%) изменений уровней TG после голодания по сравнению с исходными значениями определяли для каждой обезьяны и усредняли для каждой группы.
Величины ошибок во всех графах представлены в виде средних значений ± SEM.
ПОДРОБНОЕ ОПИСАНИЕ
Прежде, чем настоящее изобретение будет детально описано, следует понимать, что это изобретение не ограничено конкретными описанными способами и экспериментальными условиями, поскольку эти способы и условия могут варьировать. Также следует понимать, что терминология, применяемая в настоящем документе, предназначена только для описания конкретных вариантов осуществления и не предназначена для ограничения, поскольку объем настоящего изобретения ограничен только приложенной формулой изобретения.
Если не определено иначе, все технические и научные термины, применяемые в настоящем документе, имеют те же значения, что и традиционно применяемые специалистами в данной области, к которой относится это изобретение. Хотя в практическом осуществлении или исследованиях по настоящему изобретению можно использовать любые способы и материалы, сходные или эквивалентные описываемым в настоящем документе, описаны предпочтительные способы и материалы.
Определения
Как используют в настоящем документе, термин "ангиопоэтин-подобный белок 4 человека" или "hANGPTL4" относится к hANGPTL4 с последовательностью нуклеиновой кислоты SEQ ID NO:475 и аминокислотной последовательностью SEQ ID NO:476 или его биологически активному фрагменту.
Как используют в настоящем документе, термин "антитело", предназначен для обозначения молекул иммуноглобулина, состоящих из четырех полипептидных цепей, двух тяжелых (H) цепей и двух легких (L) цепей, связанных дисульфидными связями. Каждая тяжелая цепь состоит из вариабельной области тяжелой цепи (HCVR) и константной области тяжелой цепи (CH; состоит из доменов CH1, CH2 и CH3). Каждая легкая цепь состоит из вариабельной области легкой цепи (LCVR) и константной области легкой цепи (CL). HCVR и LCVR могут далее разделяться на участки гипервариабельности, которые называют определяющими комплементарность областями (CDR), перемежающиеся с более консервативными участками, которые называют каркасными областями (FR). HCVR и LCVR состоят из трех CDR и четырех FR, которые расположены от аминоконцевой области к карбоксиконцевой области в следующем порядке: FR1, CDR1, FR2, CDR2, FR3, CDR3 и FR4.
Также возможна замена одного или нескольких остатков CDR или пропуск одной или нескольких CDR. В научной литературе описаны антитела, в которых отсутствие одной или двух CDR не мешает связыванию. Padlan et al. (1995 FASEB J. 9:133-139) анализировали участки контакта между антителами и их антигенами на основе опубликованных кристаллических структур и заключили, что только приблизительно от одной пятой до одной трети остатков CDR фактически контактируют с антигеном. Padlan также обнаружил множество антител, в которых одна или две CDR не содержали аминокислот, контактирующих с антигеном (см. также Vajdos et al. 2002 J Mol Biol 320:415-428).
Остатки CDR, не контактирующие с антигеном, можно определять на основе предыдущих исследований (например, остатки H60-H65 в CDRH2 часто не являются необходимыми), исходя из областей CDR по Kabat, которые лежат вне CDR по Chothia, посредством молекулярного моделирования и/или эмпирически. Если CDR или ее остаток(и) пропущен, он, как правило, заменяется аминокислотой, занимающей соответствующую позицию в другой последовательности антитела человека или в консенсусе таких последовательностей. Позиции для замен в CDR и аминокислоты, которые могут быть замещены, также можно определять эмпирически. Эмпирические замены могут представлять собой консервативные или неконсервативные замены.
Как используют в настоящем документе, термин "антитело человека" включает антитела, содержащие вариабельные и константные области, полученные из последовательностей иммуноглобулинов зародышевой линии человека. mAb человека по изобретению могут включать аминокислотные остатки, не кодируемые последовательностями иммуноглобулинов зародышевой линии человека (например, мутации, вызванные случайным или сайт-специфическим мутагенезом in vitro или соматическим мутированием in vivo), например, в CDR и, в частности, в CDR3. Однако, как используют в настоящем документе, термин "антитело человека" не включает mAb, в которых последовательности CDR, полученные из зародышевых линий других видов млекопитающих (например, мыши), встроили к последовательностям FR человека.
Полностью антитела человека против hANGPTL4, описываемые в настоящем документе, могут содержать одну или несколько замен аминокислот, вставок и/или делеций в каркасной области и/или в CDR вариабельных доменов тяжелой и легкой цепей по сравнению с соответствующими последовательностями зародышевой линии. Такие мутации можно легко выявлять посредством сравнения аминокислотных последовательностей, описываемых в настоящем документе, с последовательностями зародышевых линий, доступными, например, в базах данных последовательностей антител. Настоящее изобретение относится к антителам и их антигенсвязывающим фрагментам, полученным из любых аминокислотных последовательностей, описываемых в настоящем документе, где одна или несколько аминокислот в составе одной или нескольких каркасных областей и/или областей CDR заменены на соответствующий остаток(и) последовательности зародышевой линии, из которой получено антитело, или на соответствующий остаток(и) другой последовательности зародышевой линии человека, или на консервативную аминокислотную замену соответствующего остатка(ов) зародышевой линии (такие изменения последовательности в настоящем документе обозначают в целом как "мутации зародышевой линии"). Специалист в данной области на основе последовательностей вариабельных областей легкой цепи и тяжелой цепи, описываемых в настоящем документе, может легко получать многочисленные антитела и антигенсвязывающие фрагменты, содержащие одну или несколько индивидуальных обратных мутаций зародышевой линии или их сочетания. В определенных вариантах осуществления все остатки каркаса и/или CDR в доменах VH и/или VL обратно мутировали с восстановлением остатков исходной последовательности зародышевой линии, из которой получено данное антитело. В других вариантах осуществления только определенные остатки обратно мутируют, восстанавливая исходную последовательность зародышевой линии, например, только мутировавшие остатки в пределах первых 8 аминокислот FR1 или в пределах последних 8 аминокислот FR4, или только мутировавшие остатки в пределах CDR1, CDR2 или CDR3. В других вариантах осуществления один или несколько остатков каркаса и/или CDR мутировали на соответствующий остаток(и) другой последовательности зародышевой линии (т.е., последовательности зародышевой линии, которая отличается от последовательности зародышевой линии, из которой исходно получено данное антитело). Кроме того, антитела по настоящему изобретению могут содержать любые комбинации двух или более мутаций зародышевой линии в каркасной области и/или области CDR, например, где определенные индивидуальные остатки мутировали на соответствующие остатки определенной последовательности зародышевой линии, в то время как другие определенные остатки, отличающиеся от исходной последовательности зародышевой линии, сохранились или мутировали на соответствующий остаток другой последовательности зародышевой линии. После получения антитела и антигенсвязывающие фрагменты, содержащие одну или несколько мутаций зародышевой линии, можно легко проверять на одно или несколько желаемых свойств, таких как повышенная специфичность связывания, повышенная аффинность связывания, повышенные или усиленные антагонистические или агонистические биологические свойства (соответственно), пониженная иммуногенность и т.д. Антитела и антигенсвязывающие фрагменты, полученные этим общим способом, включены в настоящее изобретение.
Настоящее изобретение также относится к антителам против ANGPTL4, содержащим варианты любой из аминокислотных последовательностей HCVR, LCVR и/или CDR, описываемых в настоящем документе, и содержащим одну или несколько консервативных замен. Например, настоящее изобретение относится к антителам против ANGPTL4, содержащим аминокислотные последовательности HCVR, LCVR и/или CDR с, например, 10 или менее, 8 или менее, 6 или менее, 4 или менее, 2 или 1 консервативных аминокислотных замен по сравнению с любой из аминокислотных последовательностей HCVR, LCVR и/или CDR, описываемых в настоящем документе. В одном из вариантов осуществления HCVR содержит аминокислотную последовательность SEQ ID NO:487 с 10 или менее консервативными аминокислотными заменами. В другом варианте осуществления HCVR содержит аминокислотную последовательность SEQ ID NO:487 с 8 или менее консервативными аминокислотными заменами. В другом варианте осуществления HCVR содержит аминокислотную последовательность SEQ ID NO:487 с 6 или менее консервативными аминокислотными заменами. В другом варианте осуществления HCVR содержит аминокислотную последовательность SEQ ID NO:487 с 4 или менее консервативными аминокислотными заменами. В еще одном варианте осуществления HCVR содержит аминокислотную последовательность SEQ ID NO:487 с 2 или 1 консервативными аминокислотными заменами. В одном из вариантов осуществления LCVR содержит аминокислотную последовательность SEQ ID NO:44 с 10 или менее консервативными аминокислотными заменами. В другом варианте осуществления LCVR содержит аминокислотную последовательность SEQ ID NO:44 с 8 или менее консервативными аминокислотными заменами. В другом варианте осуществления LCVR содержит аминокислотную последовательность SEQ ID NO:44 с 6 или менее консервативными аминокислотными заменами. В другом варианте осуществления LCVR содержит аминокислотную последовательность SEQ ID NO:44 с 4 или менее консервативными аминокислотными заменами. В еще одном варианте осуществления LCVR содержит аминокислотную последовательность SEQ ID NO:44 с 2 или 1 консервативными аминокислотными заменами.
Если не указано иначе, термин "антитело", как используют в настоящем документе, следует понимать как включающий молекулы антител, содержащие две тяжелые цепи иммуноглобулинов и две легкие цепи иммуноглобулинов (т.е., "полные молекулы антител"), а также их антигенсвязывающие фрагменты. Термины "антигенсвязывающий участок" антитела, "антигенсвязывающий фрагмент" антитела и т.п., как используют в настоящем документе, включают любой природный, получаемый ферментативно, синтетически или методами генетической инженерии полипептид или гликопротеин, который специфически связывается с антигеном с образованием комплекса. Антигенсвязывающие фрагменты антитела можно получать, например, из полной молекулы антитела посредством любых подходящих стандартных способов, таких как протеолитическое расщепление или способы рекомбинантной генетической инженерии, которые включают обработку и экспрессию ДНК, кодирующей вариабельные и (необязательно) константные домены антитела. Такая ДНК известна и/или легкодоступна, например, в коммерческих источниках, библиотеках ДНК (включая, например, библиотеки антител в формате фагового дисплея) или ее можно синтезировать. ДНК можно секвенировать и обрабатывать химически или посредством способов молекулярной биологии, например, для расположения одного или нескольких вариабельных и/или константных доменов в подходящем порядке, или для введения кодонов, создания остатков цистеина, модифицирования, добавления или удаления аминокислот и т.д.
Неограничивающие примеры антигенсвязывающих фрагментов включают: (i) фрагменты Fab; (ii) фрагменты F(ab')2; (iii) фрагменты Fd; (iv) фрагменты Fv; (v) одноцепочечные молекулы Fv (scFv); (vi) фрагменты dAb; и (vii) минимальные распознающие компоненты, состоящие из аминокислотных остатков, которые имитируют гипервариабельную область антитела (например, изолированная определяющая комплементарность область (CDR)). Другие молекулы, полученные способами генетической инженерии, такие как диатела, триатела, тетратела и минитела, также включены в термин "антигенсвязывающий фрагмент", как используют в настоящем документе.
Антигенсвязывающий фрагмент антитела, как правило, содержит по меньшей мере один вариабельный домен. Вариабельный домен может иметь любой размер и любую аминокислотную композицию и, как правило, содержит по меньшей мере одну CDR, прилежащую к или расположенную внутри одной или нескольких каркасных последовательностей. В антигенсвязывающих фрагментах, содержащих домен VH домен, связанный с доменом VL, домены VH и VL могут располагаться относительно друг друга любым подходящим образом. Например, вариабельная область может быть димерной и содержать димеры VH-VH, VH-VL или VL-VL. Альтернативно, антигенсвязывающий фрагмент антитела может содержать мономерный домен VH или VL.
В определенных вариантах осуществления антигенсвязывающий фрагмент антитела может содержать по меньшей мере один вариабельный домен, ковалентно связанный с по меньшей мере одним константным доменом. Неограничивающие примеры конфигураций вариабельных и константных доменов, которые можно найти в антигенсвязывающем фрагменте антитела по настоящему изобретению, включают: (i) VH-CH1; (ii) VH-CH2; (iii) VH-CH3; (iv) VH-CH1-CH2; (v) VH-CH1-CH2-CH3; (vi) VH-CH2-CH3; (vii) VH-CL; (viii) VL-CH1; (ix) VL-CH2; (x) VL-CH3; (xi) VL-CH1-CH2; (xii) VL-CH1-CH2-CH3; (xiii) VL-CH2-CH3; и (xiv) VL-CL. В любой конфигурации вариабельных и константных доменов, включая любую из конфигураций, приведенных выше в качестве примера, вариабельные и константные домены могут быть непосредственно связаны друг с другом или могут быть связаны полной или частичной шарнирной областью или линкерной областью. Шарнирная область может состоять из по меньшей мере 2 (например, 5, 10, 15, 20, 40, 60 или более) аминокислот, которые образуют подвижное или полуподвижное соединение между прилежащими вариабельными и/или константными доменами в единой молекуле полипептида. Кроме того, антигенсвязывающий фрагмент антитела по настоящему изобретению может содержать гомодимер или гетеродимер (или другой мультимер) любых конфигураций вариабельных и константных доменов, приведенных выше, нековалентно связанных друг с другом и/или с одним или несколькими мономерными доменами VH или VL (например, посредством дисульфидной связи(ей)).
Как и полные молекулы антитела, антигенсвязывающие фрагменты могут быть моноспецифическими или мультиспецифическими (например, биспецифическими). Мультиспецифический антигенсвязывающий фрагмент антитела, как правило, содержит по меньшей мере два различных вариабельных домена, где каждый вариабельный домен способен к специфическому связыванию с отдельным антигеном или с другим эпитопом на том же антигене. Любую форму мультиспецифического антитела, включая приведенные в качестве примера формы биспецифического антитела, описываемые в настоящем документе, можно применять в качестве антигенсвязывающего фрагмента антитела по настоящему изобретению с применением обычных способов, доступных в данной области.
В определенных вариантах осуществления антитело или фрагменты антител по изобретению можно объединять в терапевтическую составную группу ("иммуноконъюгат"), такую как цитотоксин, химиотерапевтическое лекарственное средство, иммуносупрессор или радиоактивный изотоп.
Термин "специфически связывается" и т.п. означает, что антитело или его антигенсвязывающий фрагмент образуют комплекс с антигеном, относительно стабильный в физиологических условиях. Специфическое связывание можно характеризовать посредством равновесной константы диссоциации (KD), которая составляет приблизительно 1×10-6 M или менее (т.е., меньшие значения KD означают более прочное связывание). Способы определения того, связываются ли специфически две молекулы, хорошо известны в данной области и включают, например, равновесный диализ, поверхностный плазмонный резонанс и т.п. Выделенное антитело, которое специфически связывается с hANGPTL4, может, однако, проявлять перекрестную реактивность по отношению к другим антигенам, таким как молекулы ANGPTL4 других видов, например, ANGPTL4 яванского макака, и/или hANGPTL3 с аминокислотной последовательностью SEQ ID NO:485. Кроме того, мультиспецифические антитела (например, биспецифические), которые связываются с hANGPTL4 и одним или несколькими дополнительными антигенами, тем не менее рассматривают как антитела, которые "специфически связываются" с hANGPTL4, как используют в настоящем документе.
Термин "высокая аффинность" антитела относится к тем антителам, для которых аффинность связывания с hANGPTL4, выраженная в виде KD, составляет приблизительно 1×10-9 M или менее, приблизительно 0,5×10-9 M или менее, приблизительно 0,25×10-9 M или менее, приблизительно 1×10-10 M или менее или приблизительно 0,5×10-10 M или менее, как показано посредством поверхностного плазмонного резонанса, например, BIACORE™ или ELISA.
Как используют в настоящем документе, термин "KD" предназначен для обозначения равновесной константы диссоциации определенного взаимодействия антитело-антиген.
Под термином "низкая скорость диссоциации", "Koff" или "kd" понимают антитело, которое диссоциирует от hANGPTL4 с константой скорости реакции, составляющей 1×10-3 с-1 или менее, предпочтительно, 1×10-4 с-1 или менее, как определяют посредством поверхностного плазмонного резонанса, например, BIACORE™.
Под термином "встроенная константа аффинности" или "ka" понимают антитело, которое ассоциируется с hANGPTL4 с константой скорости, составляющей приблизительно 1×103 M-1с-1 или более, как определяют посредством поверхностного плазмонного резонанса, например, BIACORE™.
"Выделенное антитело", как используют в настоящем документе, предназначено для обозначения антитела, которое в значительной степени свободно от других mAb с другой антигенной специфичностью (например, выделенное антитело, которое специфически связывается с hANGPTL4, в значительной степени свободно от mAb, которые специфически связывают антигены, отличные от hANGPTL4). Выделенное антитело, которое специфически связывается с hANGPTL4, может, однако, проявлять перекрестную реактивность по отношению к другим антигенам, таким как молекулы ANGPTL4 других видов, таких как яванский макак, и/или другим родственным белкам, таким как ANGPTL3 человека.
"Нейтрализующее антитело", как используют в настоящем документе (или "антитело, которое нейтрализует активность ANGPTL4"), предназначено для обозначения антитела, связывание которого с ANGPTL4 приводит к ингибированию по меньшей мере одной биологической активности ANGPTL4. Это ингибирование биологической активности ANGPTL4 можно оценивать посредством оценки одного или нескольких индикаторов биологической активности ANGPTL4 одним или несколькими из стандартных анализов in vitro или in vivo, известных в данной области (также см. примеры ниже).
Как используют в настоящем документе, термин "поверхностный плазмонный резонанс" относится к оптическому феномену, который позволяет проводить анализ биоспецифичных взаимодействий в реальном времени посредством регистрации изменений концентрации белков в биосенсорной матрице, например, с использованием системы BIACORE™ (Pharmacia Biosensor AB, Uppsala, Sweden и Piscataway, N.J.).
Термин "эпитоп" обозначает участок антигена, который связывается антителом. Эпитопы можно определять как структурные или функциональные. Функциональные эпитопы, как правило, представляют собой подгруппы структурных эпитопов и содержат остатки, которые непосредственно определяют аффинность взаимодействия. Эпитопы также могут быть конформационными, то есть состоящими из нелинейных аминокислот. В определенных вариантах осуществления эпитопы могут включать детерминанты, которые представляют собой химически активные поверхностные группы молекул, таких как аминокислоты, боковые цепи сахаров, фосфорильные группы или сульфонильные группы и, в определенных вариантах осуществления, могут иметь трехмерные структурные характеристики и/или специфичные зарядные характеристики.
Термин "существенная идентичность" или "в значительной степени идентичные" по отношению к нуклеиновой кислоте или ее фрагменту означают, что при оптимальном выравнивании с учетом соответствующих нуклеотидных вставок или делеций относительно другой нуклеиновой кислоты (или ее комплементарной цепи) обнаруживается идентичность нуклеотидных последовательностей, составляющая по меньшей мере приблизительно 90% и, более предпочтительно, по меньшей мере приблизительно 95%, 96%, 97%, 98% или 99% нуклеотидных остатков, как определяют посредством любого хорошо известного способа определения идентичности последовательностей, такого как FASTA, BLAST или GAP, как описано ниже.
В применении к полипептидам термин "существенное сходство" или "в значительной степени сходные" означает, что две последовательности пептида при оптимальном выравнивании, таком как выравнивание посредством программ GAP или BESTFIT с использованием установленного по умолчанию веса пропусков, обнаруживают идентичность последовательностей, составляющую по меньшей мере 90%, даже более предпочтительно, по меньшей мере 95%, 98% или 99%. Предпочтительно, позиции неидентичных остатков отличаются друг от друга консервативными аминокислотными заменами. "Консервативная аминокислотная замена" представляет собой замену, при которой аминокислотный остаток заменяется другим аминокислотным остатком, боковая цепь (R-группа) которого имеет сходные химические свойства (например, заряд или гидрофобность). Как правило, консервативная аминокислотная замена по существу не меняет функциональные свойства белка. В тех случаях, когда две или более аминокислотных последовательностей отличаются друг от друга консервативными заменами, процент или степень сходства можно повышать, делая поправку на консервативность замены. Способы проведения таких оценок хорошо известны специалистам в данной области. См., например, Pearson (1994) Methods Mol. Biol. 24: 307-331. Примеры групп аминокислот, которые содержат боковые цепи со сходными химическими свойствами, включают 1) алифатические боковые цепи: глицин, аланин, валин, лейцин и изолейцин; 2) алифатические гидроксильные боковые цепи: серин и треонин; 3) амид-содержащие боковые цепи: аспарагин и глутамин; 4) ароматические боковые цепи: фенилаланин, тирозин, и триптофан; 5) основные боковые цепи: лизин, аргинин, и гистидин; 6) кислотные боковые цепи: аспартат и глутаминат, и 7) сера-содержащие боковые цепи: цистеин и метионин. Предпочтительные группы консервативных аминокислотных замен следующие: валин-лейцин-изолейцин, фенилаланин-тирозин, лизин-аргинин, аланин-валин, глутаминат-аспартат и аспарагин-глутамин. Альтернативно, консервативная замена представляет собой любое изменение, имеющее положительное значение на матрице логарифмического правдоподобия PAM250, описанной в Gonnet et al. (1992) Science 256: 1443 45. "Умеренно консервативная" замена представляет собой любое изменение, имеющее не отрицательное значение в матрице логарифмического правдоподобия PAM250.
Сходство последовательностей для полипептидов, как правило, измеряют с применением программного обеспечения для анализа последовательностей. Программное обеспечение для анализа белков сравнивает сходные последовательности с использованием оценок сходства, присвоенных различным заменам, делециям и другим модификациям, включая консервативные аминокислотные замены. Например, программное обеспечение GCG содержит программы, такие как GAP и BESTFIT, которые можно использовать с установленными по умолчанию параметрами для определения гомологичности последовательностей или идентичности последовательностей между двумя близкородственными полипептидами, такими как гомологичные полипептиды разных видов организмов, или между белком дикого типа и его мутеином. См., например, GCG Version 6,1. Последовательности полипептидов также можно сравнивать с использованием FASTA с установленными по умолчанию или рекомендуемыми параметрами; программа в составе GCG Version 6,1. FASTA (например, FASTA2 и FASTA3) предоставляет выравнивания и проценты идентичности последовательностей для участков наилучшего перекрывания заданной и искомой последовательности (Pearson (2000) выше). Другие предпочтительные алгоритмы для сравнения последовательностей по изобретению с последовательностями из базы данных, содержащей большое количество последовательностей различных организмов, включают компьютерную программу BLAST, в частности, BLASTP или TBLASTN, в которой используют установленные по умолчанию параметры. См., например, Altschul et al. (1990) J. Mol. Biol. 215: 403 410 и (1997) Nucleic Acids Res. 25:3389 402.
Под фразой "терапевтически эффективное количество" понимают количество, которое оказывает желаемое действие, для которого его вводят. Точное количество зависит от цели лечения, возраста и размера тела подлежащего лечению индивидуума, способа введения и т.п., и может быть установлено специалистом в данной области с применением известных способов (см., например, Lloyd (1999) The Art, Science and Technology of Pharmaceutical Compounding).
Получение антител человека
Способы получения антител человека у трансгенных мышей известны в данной области. Любые известные способы можно использовать в контексте настоящего изобретения для получения антител человека, которые специфично связываются с ANGPTL4.
С применением VELOCIMMUNE™ или любого другого известного способа получения моноклональных антител, химерные антитела против ANGPTL4 с высокой аффинностью сначала изолируют с сохранением вариабельной области человека и константной области мыши. Как указано в экспериментальном разделе ниже, антитела характеризуют и выбирают на основе желаемых характеристик, включая аффинность, селективность, эпитоп и т.п.
Как правило, антитела по настоящему изобретению обладают очень высокой аффинностью, как правило, проявляя KD, составляющую приблизительно от 10-12 M до приблизительно 10-9 M, измеренную при связывании с антигеном, иммобилизированным либо на твердой фазе, либо в жидкой фазе. Константные области мыши заменяют желаемыми константными областями человека, например, дикого типа IgG1 (SEQ ID NO:481) или IgG4 (SEQ ID NO:482), или модифицированными IgG1 или IgG4 (например, SEQ ID NO:483) с получением полностью антител человека по изобретению. В то время как выбранная константная область может варьировать в зависимости от конкретного применения, высокоаффинные антигенсвязывающие и специфичные характеристики антител сохраняются в вариабельной области.
Картирование эпитопов и связанные способы
Для поиска антител, которые связываются с определенным эпитопом, можно проводить общепринятый перекрестный конкурентный анализ, такой как описанный в Antibodies, Harlow и Lane (Cold Spring Harbor Press, Cold Spring Harb., NY). Другие способы включают сканирующий аланином анализ мутантов, блот-анализ пептидов (Reineke (2004) Methods Mol Biol 248:443-63), или анализ с использованием расщепления пептидов. Кроме того, можно использовать способы, такие как вырезание эпитопа, выделение эпитопа и химическая модификация антигенов (Tomer (2000) Protein Science 9: 487-496).
Термин "эпитоп" относится к участку на антигене, на который отвечают B-клетки и/или T-клетки. Эпитопы для B-клеток могут быть образованы как смежными аминокислотами, так и несмежными аминокислотами, которые сближаются третичной укладкой белка. Эпитопы, образованные смежными аминокислотами, как правило, сохраняются при воздействии денатурирующих растворителей, в то время как эпитопы, образованные третичной укладкой, как правило, утрачиваются при обработке денатурирующими растворителями. Эпитоп, как правило, включает по меньшей мере 3 и более, как правило, по меньшей мере 5 или 8-10 аминокислот с уникальной пространственной конформацией.
Определение профиля с помощью модификации (MAP), также известное как определение профиля антител на основе структуры антигена (ASAP), представляет собой метод классификации большого числа моноклональных антител (mAb) к одному и тому же антигену в соответствии со сходствами профиля связывания каждого антитела с химически или энзиматически модифицированными антигенными поверхностями (США 2004/0101920). Каждая категория может отражать уникальный эпитоп, который либо четко отличается от эпитопа, представленного в любой другой категории, либо частично перекрывается с ним. Подобная технология позволяет быстро отделять генетически идентичные антитела, так что их идентификация может быть сконцентрирована на генетически отличающихся антителах. В случае использования для скрининга гибридомы MAP может способствовать идентификации редких клонов гибридомы, производящих mAb с нужными характеристиками. MAP можно применять для сортировки анти-ANGPTL4 mAb по изобретению на группы mAb, связывающих различные эпитопы.
ANGPTL4 содержит аминоконцевой суперспиральный домен и карбоксиконцевой фибриногеноподобный домен, и полноразмерный белок ANGPTL4 образует олигомер, который поддерживается межмолекулярными дисульфидными связями (Ge et al., 2004, J Bio Chem 279(3):2038-2045). Опубликовано, что N-концевой суперспиральный домен опосредует олигомеризацию ANGPTL4 (Ge et al., выше) и также важен для ингибирования активности LPL (Ge et al., 2005, J Lipid Res 46:1484-1490; и Ono et al., 2003, J Biol Chem 278:41804-41809). Таким образом, в определенных вариантах осуществления антитело против hANGPTL4 или антигенсвязывающий фрагмент антитела связывают эпитоп в N-концевом суперспиральном домене (остатки 1-123) hANGPTL4 (SEQ ID NO:476). В определенных вариантах осуществления антитело против hANGPTL4 или его фрагмент связывают эпитоп в участке приблизительно от остатка 1 до приблизительно остатка 25, приблизительно от остатка 25 до приблизительно остатка 50, приблизительно от остатка 50 до приблизительно остатка 75, приблизительно от остатка 75 до приблизительно остатка 100, приблизительно от остатка 100 до приблизительно остатка 125, приблизительно от остатка 125 до приблизительно остатка 150 hANGPTL4 (SEQ ID NO:476). В некоторых вариантах осуществления антитело или фрагмент антитела связывают эпитоп, который включает более одного указанных эпитопов в N-концевом суперспиральном домене hANGPTL4. В других вариантах осуществления антитело hANGPTL4 или его фрагмент связывают один или несколько фрагментов hANGPTL4, например, фрагмент от остатка 26 до остатка 406, от остатка 26 до остатка 148, от остатка 34 до остатка 66 и/или от остатка 165 до остатка 406 SEQ ID NO:476.
Настоящее изобретение относится к антителам hANGPTL4, которые связываются с тем же эпитопом, что и любое из определенных антител, приведенных в качестве примера в настоящем документе. Аналогично, настоящее изобретение также относится к антителам против hANGPTL4, которые конкурируют за связывание с фрагментом hANGPTL4 или hANGPTL4 с любым из определенных антител, приведенных в качестве примера в настоящем документе.
Посредством применения общепринятых способов, известных в данной области, можно легко определять, связывается ли антитело с тем же эпитопом, что и исходное антитело против hANGPTL4, или конкурирует с ним. Например, для определения того, связывается ли изучаемое антитело с тем же эпитопом, что и исходное антитело к hANGPTL4 по изобретению, исходному антителу позволяют связаться с белком или пептидов hANGPTL4 в условиях насыщения. Далее оценивают способность изучаемого антитела связываться с молекулой hANGPTL4. Если изучаемое антитело способно связаться с hANGPTL4 после насыщения соединения с исходным антителом против hANGPTL4, можно заключить, что изучаемое антитело связывается с другим эпитопом по сравнению с исходным антителом против hANGPTL4. С другой стороны, если изучаемое антитело не способно связаться с молекулой hANGPTL4 после насыщения соединения исходного антитела против hANGPTL4, можно заключаить, что изучаемое антитело может связываться с тем же эпитопом, что и исходное антитело против hANGPTL4 по изобретению.
Для определения того, конкурирует ли антитело за связывание с исходным антителом к hANGPTL4, описанный выше способ связывания проводят в двух направлениях: в первом направлении исходному антителу позволяют связаться с молекулой hANGPTL4 в условиях насыщения с последующей оценкой связывания изучаемого антитела с молекулой hANGPTL4. Во втором направлении изучаемому антителу позволяют связаться с молекулой hANGPTL4 в условиях насыщения с последующей оценкой связывания исходного антитела с молекулой ANGPTL4. Если при обоих направлениях только первое (насыщающее) антитело способно связываться с молекулой ANGPTL4, заключают, что изучаемое антитело и исходное антитело конкурируют за связывание с hANGPTL4. Как будет понятно специалистам в данной области, антитело, которое конкурирует за связывание с исходным антителом, может необязательно связываться с тем же эпитопом, то и исходное антитело, но может и стерически блокировать связывание исходного антитела посредством связывания перекрывающегося или прилежащего эпитопа.
Два антитела связываются с одним и тем же эпитопом или перекрывающимися эпитопами, если каждый из них конкурентно ингибирует (блокирует) связывание другого с антигеном. То есть, 1-, 5-, 10-, 20- или 100-кратный избыток одного антитела ингибирует связывание другого на по меньшей мере 50%, но предпочтительно, 75%, 90% или даже 99%, как показано с использованием анализа конкурентного связывания (см., например, Junghans et al., Cancer Res, 1990:50:1495-1502). Альтернативно, два антитела содержат одинаковый эпитоп, если почти все мутации аминокислот в антигене, которые сокращают или подавляют связывание одного антитела, сокращают или подавляют связывание другого. Два антитела содержат перекрывающиеся эпитопы, если некоторые мутации аминокислот, которые сокращают или подавляют связывание одного антитела, сокращают или подавляют связывание другого.
Дополнительное общепринятое экспериментирование (например, анализ пептидных мутаций и связывания) можно затем проводить для подтверждения того, что наблюдаемое отсутствие связывания изучаемого антитела действительно вызвано связыванием с тем же эпитопом, что и у исходного антитела, или что стерическая блокировка (или другой феномен) вызывает отсутствие наблюдаемого связывания. Эксперименты такого рода можно проводить с применением ELISA, RIA, поверхностного плазмонного резонанса, проточной цитометрии или любых других качественных или количественных анализов связывания антитела, доступных в данной области.
Иммуноконъюгаты
Настоящее изобретение относится к анти-ANGPTL4 моноклональному антителу человека, конъюгированному с терапевтической составной группой ("иммуноконъюгат"), такой как цитотоксин, химиотерапевтическое лекарственное средство, иммуносупрессор или радиоактивный изотоп. Цитотоксические средства включают любые средства, пагубные для клеток. Примеры подходящих цитотоксических средств и химиотерапевтических средств для получения иммуноконъюгатов известны в данной области, см. например, WO 05/103081.
Биспецифические антитела
Антитела по настоящему изобретению могут быть моноспецифическими, биспецифическими или мультиспецифическими. Мультиспецифические mAb могут быть специфичными к различным эпитопам одного полипептида-мишени или могут содержать антигенсвязывающие домены, специфичные к более чем одному полипептиду-мишени. См., например, Tutt et al. (1991) J. Immunol. 147:60-69. Человеческие анти-hANGPTL4 mAb могут быть связаны или могут коэкспрессироваться с другой функциональной молекулой, например, с другим пептидом или белком. Например, антитело или его фрагмент можно функционально связывать (например, посредством химического сопряжения, генной гибридизации, нековалентного связывания или других способов) с одной или несколькими другими молекулярными структурами, такими как другое антитело или фрагмент антитела, с получением биспецифического или полиспецифического антитела со второй специфичностью связывания.
Примеры структуры биспецифичесихих антител, которые можно использовать в контексте настоящего изобретения, подразумевают применение первого CH3-домена иммуноглобулина (Ig) и второго CH3-домена Ig, где первый и второй CH3-домены Ig отличаются друг от друга по меньшей мере одной аминокислотой, и где по меньшей мере одно аминокислотное различие сокращает связывание биспецифического антитела с Белком A по сравнению с биспецифическим антителом, не несущим аминокислотное различие. В одном из вариантов осуществления первый CH3-домен Ig связывает Белок A, а второй CH3-домен Ig содержит мутацию, которая сокращает или подавляет связывание Белка A, такую как модификация H95R (согласно нумерации экзонов IMGT; H435R согласно нумерации EU). Второй CH3 может дополнительно содержать модификацию Y96F (согласно IMGT; Y436F согласно EU). Дальнейшие модификации, которые можно найти во втором CH3, включают: D16E, L18M, N44S, K52N, V57M и V82I (согласно IMGT; D356E, L358M, N384S, K392N, V397M и V422I согласно EU) в случае антитела IgG1; N44S, K52N и V82I (IMGT; N384S, K392N и V422I согласно EU) в случае антитела IgG2; и Q15R, N44S, K52N, V57M, R69K, E79Q и V82I (согласно IMGT; Q355R, N384S, K392N, V397M, R409K, E419Q и V422I согласно EU) в случае антитела IgG4. Вариации структуры биспецифического антитела, описанные выше, включены в объем настоящего изобретения.
Биоэквиваленты
Антитела против hANGPTL4 и фрагменты антител по настоящему изобретению включают белки, аминокислотные последовательности которых отличаются от аминокислотных последовательностей описанных mAb, но сохраняют способность связываться с ANGPTL4 человека. Такие варианты mAb и фрагментов антител содержат одну или несколько вставок, делеций или замен аминокислот по сравнению с исходной последовательностью, но проявляют биологическую активность, по существу эквивалентную биологической активности описанных mAb. Так же, последовательности ДНК, кодирующие антитело против hANGPTL4 по настоящему изобретению, включают последовательности, которые содержат одну или несколько вставок, делеций или замен нуклеотидов по сравнению с раскрытой последовательностью, но которые кодируют антитело против hANGPTL4 или фрагмент антитела, по существу биоэквивалентные антителу против hANGPTL4 или фрагменту антитела по изобретению. Примеры таких вариантов аминокислотных последовательностей и последовательностей ДНК описаны выше.
Два антиген-связывающих белка, или антитела, считают биоэквивалентами, если, например, они представляют собой фармацевтические эквиваленты или фармацевтические альтернативы, скорость и степень абсорбции которых не демонстрирует значимых отличий при введении в одинаковых молярных дозах при одинаковых экспериментальных условиях, как при однократном, так и при многократном введении. Некоторые антитела считают эквивалентами или фармацевтическими альтернативами, если они эквиваленты по степени абсорбции, но не по скорости абсорбции; при этом их считают биоэквивалентами, поскольку такие различия в скорости абсорбции являются преднамеренными и указаны на этикетках и не являются существенными для достижения эффективной концентрации средства в организме, например, при постоянном применении, и их считают незначимыми с медицинской точки зрения в отношении определенного изучаемого лекарственного средства. В одном из вариантов осуществления два антиген-связывающих белка представляют собой биоэквиваленты, если не наблюдается клинически значимых различий в их безопасности, чистоте и активности.
В одном из вариантов осуществления два антиген-связывающих белка представляют собой биоэквиваленты, если пациента можно один или несколько раз переводить с исходного средства на биологическое средство и наоборот без ожидаемого повышения риска неблагоприятных воздействий, включая клинически значимые изменения иммуногенности, или уменьшения эффективности, по сравнению с продолжительным лечением без такого перевода.
В одном из вариантов осуществления два антиген-связывающих белка представляют собой биоэквиваленты, если они оба действуют посредством сходного механизма или механизмов действия в условии или условиях применения, в той степени, в которой такие механизмы известны.
Биоэквивалентность можно обнаруживать способами in vivo и in vitro. Измерения биоэквивалентности включают, например, (a) тест in vivo для людей или других млекопитающих, в котором измеряют концентрацию антитела или его метаболитов в крови, плазме, сыворотке или других биологических жидкостях как функцию времени; (b) тест in vitro, который коррелирует с и является достаточно предсказуемым в отношении данных биодоступности in vivo для человека; (c) тест in vivo для людей или других млекопитающих, в котором соответствующее немедленное фармакологическое действие антитела (или его мишени) измеряют как функцию времени; и (d) контролируемое клиническое испытание, которое позволяет определять безопасность, эффективность или биодоступность или биоэквивалентность антитела.
Биоэквивалентные варианты антител против hANGPTL4 по изобретению можно получать посредством, например, получения различных замен остатков или последовательностей или удаления концевых или внутренних остатков или последовательностей, не являющихся необходимыми для биологической активности. Например, остатки цистеина, несущественные для биологической активности, можно удалять или заменять другими аминокислотами для превращения образования ненужных или неправильных внутримолекулярных дисульфидных мостиков при ренатурации.
Терапевтическое введение и терапевтические составы
Изобретение относится к терапевтическим композициям, содержащим антитела против hANGPTL4 или их антигенсвязывающие фрагменты по настоящему изобретению, и терапевтическим способам их применения. Введение терапевтических композиций согласно изобретению проводят с использованием подходящих носителей, эксципиентов и других агентов, которые включают в составы для предоставления улучшенного транспорта, доставки, переносимости и т.п. Большое количество подходящих составов можно найти в фармакологическом справочнике, известном всем химикам-фармацевтам: Remington's Pharmaceutical Sciences, Mack Publishing Company, Easton, PA. Эти составы включают, например, порошки, пасты, мази, гели, воска, масла, жиры, липид (катионный или анионный)-содержащие носители (такие как ЛИПОФЕКТИН™), конъюгаты ДНК, безводные абсорбирующие пасты, эмульсии масло-в-воде и вода-в-масле, эмульсии типа карбовакс (полиэтиленгликоли с различной молекулярной массой), полужидкие гели и полужидкие смеси, содержащие кабовакс. См. также Powell et al. "Compendium of excipients for parenteral formulations" PDA (1998) J Pharm Sci Technol 52:238-311.
Дозы могут варьировать в зависимости от возраста и размера тела субъекта, которому доставляют средство, подлежащего лечению заболевания, цели лечения, состояния, способа введения и т.п. Если антитело по настоящему изобретению применяют для лечения различных состояний и заболеваний, напрямую или косвенно связанных с ANGPTL4, включая гиперхолестеринемию, нарушений, связанных с LDL и аполипопротеином B, и нарушений метаболизма липидов и т.п., у взрослых пациентов, предпочтительно, вводить антитело по настоящему изобретению внутривенно или подкожно в однократной дозе, составляющей приблизительно от 0,01 до приблизительно 20 мг/кг массы тела, более предпочтительно, приблизительно от 0,02 до приблизительно 7, приблизительно от 0,03 до приблизительно 5 или приблизительно от 0,05 до приблизительно 3 мг/кг массы тела. В зависимости от тяжести состояния устанавливают частоту и продолжительность лечения. В определенных вариантах осуществления антитело или его антигенсвязывающий фрагмент по изобретению можно вводить в начальной дозе, составляющей по меньшей мере приблизительно от 0,1 мг до приблизительно 800 мг, приблизительно от 1 до приблизительно 500 мг, приблизительно от 5 до приблизительно 300 мг, или приблизительно от 10 до приблизительно от 200 мг, до приблизительно 100 мг или до приблизительно 50 мг. В определенных вариантах осуществления после введения начальной дозы можно вводить вторую дозу или несколько последовательных дох антитела или его антигенсвязывающего фрагмента в количестве, которое может быть приблизительно равным начальной дозе или быть меньше ее, где последующие дозы вводят с промежутком, составляющим, по меньшей мере от 1 суток до 3 суток; по меньшей мере одну неделю, по меньшей мере 2 недели; по меньшей мере 3 недели; по меньшей мере 4 недели; по меньшей мере 5 недель; по меньшей мере 6 недель; по меньшей мере 7 недель; по меньшей мере 8 недель; по меньшей мере 9 недель; по меньшей мере 10 недель; по меньшей мере 12 недель; или, по меньшей мере 14 недель.
Известны различные системы доставки, которые можно использовать для введения фармацевтической композиции по изобретению, например, заключение в липосомы, микрочастицы, микрокапсулы, рекомбинантные клетки, способные экспрессировать мутантные вирусы, опосредованный рецепторами эндоцитоз (см., например, Wu et al. (1987) J. Biol. Chem. 262:4429-4432). Способы введения в качестве неограничивающих примеров включают интрадермальный, внутримышечный, интраперитонеальный, внутривенный, подкожный, интраназальный, эпидуральный и пероральный способы введения. Композиции можно вводить посредством любого подходящего способа введения, например, посредством вливания или болюсной инъекции, абсорбции через эпителиальные или кожно-слизистые оболочки (например, слизистую оболочку ротовой полости, слизистую оболочку кишечника и прямой кишки и т.д.), и их можно вводить вместе с другими биологически активными средствами. Введение может быть системным или местным.
Фармацевтическую композицию также можно доставлять в носителе, в частности, в липосоме (см. Langer (1990) Science 249:1527-1533; Treat et al. (1989) in Liposomes in the Therapy of Infectious Disease and Cancer, Lopez Berestein и Fidler (eds.), Liss, New York, pp. 353-365; Lopez-Berestein, там же, pp. 317-327; см. там же).
В определенных ситуациях фармацевтическую композицию можно доставлять в системе контролируемого высвобождения. В одном из вариантов осуществления можно использовать помпу (см. Langer, выше; Sefton (1987) CRC Crit. Ref. Biomed. Eng. 14:201). В другом варианте осуществления можно использовать полимерные материалы; см. Medical Applications of Controlled Release, Langer и Wise (eds.), CRC Pres., Boca Raton, Florida (1974). В еще одном варианте осуществления систему контролируемого высвобождения можно помещать вблизи мишени композиции, что обеспечивает применения только части системной дозы (см., например, Goodson, in Medical Applications of Controlled Release, выше, vol. 2, pp. 115-138, 1984).
Инъецируемые препараты могут включать лекарственные формы для внутривенной, подкожной, внутрикожной и внутримышечной инъекций, вливания посредством капельницы и т.д. Эти инъецируемые препараты можно получать широко известными способами. Например, инъецируемые препараты можно получать растворением, суспендированием или эмульгированием антитела или его соли, описанной выше, в стерильной водной среде или масляной среде, традиционно применяемых для инъекций. Водные среды для инъекций представляют собой, например, физиологический раствор, изотонический раствор, содержащий глюкозу и другие вспомогательные средства, и т.д., которые можно использовать в комбинации с подходящим солюбилизатором, таким как спирт (например, этанол), полиалкоголь (например, пропиленгликоль, полиэтиленгликоль), неионное поверхностно-активное вещество [например, полисорбат 80, HCO-50 (полиоксиэтилен (50 моль) аддукт гидрогенизированного касторового масла)] и т.д. Масляные среды представляют собой, например, сезамовое масло, соевое масло и т.д., которые можно использовать в комбинации с солюбилизатором, таким как бензилбензоат, бензиловый спирт и т.д. Инъекцию, полученную таким образом, предпочтительно, вносят в подходящую ампулу. Фармацевтическую композицию по настоящему изобретению можно доставлять подкожно или внутривенно с применением стандартной иглы и шприца. Кроме того, в отношении подкожной доставки, для доставки фармацевтической композиции по настоящему изобретению можно легко применять шприц-ручку. Такое средство доставки может представлять собой средство одноразового или многоразового применения. Для многоразового шприца-ручки, как правило, применяют сменный картридж, который содержит фармацевтическую композицию. После доставки всего объема фармацевтической композиции в картридже и опустошения картриджа пустой картридж можно легко снимать и заменять новым картриджем, который содержит фармацевтическую композицию. Шприц-ручку можно применять многократно. Одноразовый шприц-ручка не содержит сменного картриджа. Вместо этого одноразовый шприц-ручку заранее заполняют фармацевтической композицией, которая хранится в резервуаре внутри устройства. После использования фармацевтической композиции в резервуаре все устройство можно выкидывать.
В подкожной доставке фармацевтической композиции по настоящему изобретению применяют многочисленные многоразовые шприцы-ручки и автоинъекторы. Неограничивающие примеры включают AUTOPEN™ (Owen Mumford, Inc., Woodstock, UK), шприц-ручку DISETRONIC™ (Disetronic Medical Systems, Burghdorf, Switzerland), шприц-ручку HUMALOG MIX 75/25™, шприц-ручку HUMALOG™, шприц-ручку HUMALIN 70/30™ (Eli Lilly и Co., Indianapolis, IN), NOVOPEN™ I, II и III (Novo Nordisk, Copenhagen, Denmark), NOVOPEN JUNIOR™ (Novo Nordisk, Copenhagen, Denmark), шприц-ручку BD™ (Becton Dickinson, Franklin Lakes, NJ), OPTIPEN™, OPTIPEN PRO™, OPTIPEN STARLET™ и OPTICLIK™ (sanofi-aventis, Frankfurt, Germany) и многие другие. Примеры одноразовых шприцев-ручек, применяемых в подкожной доставке фармацевтической композиции по настоящему изобретению, в качестве неограничивающих примеров включают шприц-ручку SOLOSTAR™ (sanofi-aventis), FLEXPEN™ (Novo Nordisk) и KWIKPEN™ (Eli Lilly).
Предпочтительно, фармацевтические композиции для перорального или парентерального применения, описанные выше, получают в виде лекарственных форм со стандартной дозой, приведенной в соответствие с дозой активных ингредиентов. Такие лекарственные формы со стандартной дозой включают, например, таблетки, пилюли, капсулы, инъекции (ампулы), суппозитории и т.д. Количество содержащегося в них указанного выше антитела, как правило, составляет приблизительно от 0,1 до приблизительно 800 мг на лекарственную форму со стандартной дозой; особенно в форме инъекции содержание указанного выше антитела составляет приблизительно от 1 до приблизительно 500 мг, приблизительно от 5 до 300 мг, приблизительно от 8 до 200 мг, и приблизительно от 10 до приблизительно 100 мг для других лекарственных форм.
Способы комбинированного лечения
Изобретение дополнительно относится к терапевтическим способам лечения заболевания или нарушения, которые напрямую или косвенно связаны с hANGPTL4, посредством введения антитела к hANGPTL4 или его фрагмента по изобретению в комбинации с одним или несколькими дополнительными терапевтическими средствами. Дополнительное терапевтическое средство может представлять собой одно или несколько средств, которые, предпочтительно, комбинируют с антителом или его фрагментом по изобретению, включая ингибиторы HMG-CoA-редуктазы, такие как церовастатин, аторвастатин, симвастатин, питавастин, розувастатин, флувастатин, ловастатин, правастатин и т.п.; ниацин; различные фибраты, такие как фенофибрат, безафибрат, ципрофибрат, клофибрат, гемфиброзил и т.п.; активаторы фактора транскрипции LXR и т.п. Кроме того, антитело к hANGPTL4 или его фрагмент по изобретению можно вводить совместно с другими ингибиторами ANGPTL4, а также ингибиторами других молекул, таких как ANGPTL3, ANGPTL5, ANGPTL6 и пропротеин конвертаза субтилизин/кексин типа 9 (PCSK9), которые вовлечены в метаболизм липидов, в частности, в гомеостаз холестерина и/или триглицеридов. Ингибиторы этих молекул включают низкомолекулярные соединения и антитела, которые специфически связываются с этими молекулами и блокируют их активность (см., например, анти-PCSK9 антитела, раскрытые в U.S. 2010/0166768 A1).
Кроме того, дополнительное терапевтическое средство может представлять собой одно или несколько противоопухолевых средств, таких как химиотерапевтические средства, антиангиогенные средства, ингибирующие рост средства, цитотоксические средства, апоптические средства и другие средства, хорошо известные в данной области для лечения злокачественной опухоли или других пролиферативных заболеваний или нарушений. Примеры противораковых средств в качестве неограничивающих примеров включают антимитотические средства, такие как доцетаксел, паклитаксел и т.п.; основанные на платине химиотерапевтические соединения, такие как цисплатин, карбоплатин, ипроплатин, оксалиплатин и т.п.; или другие общепринятые цитотоксические средства, такие как 5-фторурацил, капецитабин, иринотекан, лейковорин, гемцитабин и т.п., и антиангиогенные средства, включая антагонисты фактора роста эндотелия сосудов (VEGF), такие как антитела к VEGF, например, бевацизумаб (АВАСТИН®, Genentech) и рецептор-опосредованный блокатор VEGF, например, "VEGF trap", описанный в патенте США 7070959, антагонисты дельта-подобного лиганда 4 (DII4), такие как антитела к DII4, как описано в публикации патентной заявки США 2008/0181899, и слитый белок, содержащий внеклеточный домен DII4, например, DII4-Fc, как описано в публикации патентной заявки США 2008/0107648; ингибиторы рецепторов тирозинкиназы и/или ангиогенеза, включая сорафениб (NEXAVAR® от Bayer Pharmaceutical Corp.), сунитиниб (SUTENT® от Pfizer), пазопаниб (VOTRIENT™ от GlaxoSmithKline), тоцераниб (PALLADIA™ от Pfizer), вандетаниб (ZACTIMA™ от AstraZeneca), цедираниб (RECENTIN® от AstraZeneca), регорафениб (BAY 73-4506 от Bayer), акситиниб (AG013736 от Pfizer), лестауртиниб (CEP-701 от Cephalon), эрлотиниб (ТАРЦЕВА® от Genentech), гефитиниб (IRESSA™ от AstraZeneca), BIBW 2992 (TOVOK™ от Boehringer Ingelheim), лапатиниб (ТАЙКЕРБ® от GlaxoSmithKline), нератиниб (HKI-272 от Wyeth/Pfizer) и т.п., и фармацевтически приемлемые соли, кислоты или производные любых перечисленных выше средств. Кроме того, другие терапевтические средства, такие как анальгетики, противовоспалительные средства, включая нестероидные противовоспалительные лекарственные средства (NSAIDS), такие как ингибиторы Cox-2 и т.п., также можно вводить совместно с антителом против hANGPTL4 или его фрагментом по изобретению с тем, чтобы облегчить и/или сократить симптомы, сопутствующие злокачественной опухоли.
Антитело против hANGPTL4 или его фрагмент по изобретению и дополнительное терапевтическое средство(а) можно вводить вместе или раздельно. При применении раздельных лекарственных форм антитело или его фрагмент по изобретению и дополнительные средства можно вводить одновременно или раздельно в разное время, т.е., последовательно, в подходящем порядке.
Диагностическое применение антител
Антитела против ANGPTL4 по настоящему изобретению можно также применять для обнаружения и/или измерения ANGPTL4 в образце, например, для диагностических целей. Например, анти-ANGPTL4 Ab или его фрагмент можно использовать для диагностики состояния или заболевания, которое характеризуется нарушенной экспрессией (например, избыточная экспрессия, недостаточная экспрессия, отсутствие экспрессии и т.д.) ANGPTL4. Примеры диагностических анализов для ANGPTL4 могут включать, например, взаимодействие образца, полученного от пациента, с анти-ANGPTL4 Ab по изобретению, где антитело к ANGPTL4 помечено детектируемой меткой или репортерной молекулой или применяется для селективного захвата и изоляции белка ANGPTL4 из образца пациента. Альтернативно немеченые анти-ANGPTL4 Ab можно использовать в диагностических целях в комбинации с вторичным антителом, которое помечено меткой. Детектируемая метка или репортерная молекула могут представлять собой радиоактивный изотоп, такой как 3H, 14C, 32P, 35S, 131I или 125I; флуоресцентную или хемилюминесцентную составную группу, такую как флуоресцеинизотиоцианат или родамин; или фермент, такой как щелочная фосфатаза, β-галактозидаза, пероксидаза хрена или люцифераза. Анализы, которые можно использовать для обнаружения или измерения ANGPTL4 в образце, включают твердофазный иммуноферментный анализ (ELISA), радиоиммунологический анализ (RIA), активируемую флуоресценцией сортировку клеток (FACS) и т.п.
ПРИМЕРЫ
Следующие примеры приведены для предоставления специалистам в данной области полного раскрытия и описания того, как получать и применять композиции и способы по изобретению, и не предназначены для ограничения объема документа, который авторы изобретения относят к своему изобретению. Были приняты меры для обеспечения точности в отношении применяемых количественных значений, но следует учитывать некоторые экспериментальные ошибки и отклонения. Если не указано иначе, молекулярная масса представляет собой среднюю молекулярную массу, температура выражена в градусах Цельсия, а давление равно или приблизительно равно атмосферному.
Пример 1: Получение антител человека против ANGPTL4 человека.
Мышей VELOCIMMUNE™ иммунизировали ANGPTL4 человека, и иммунный ответ антител отслеживали посредством антиген-специфического иммунологического анализа с применением сыворотки, полученной от этих мышей. B-клетки, экспрессирующие анти-hANGPTL4, собирали из селезенок иммунизированных мышей, для которых были показаны повышенные титры антител к hANGPTL4, и сливали с миеломными клетками мыши с образованием гибридом. Гибридомы скринировали и отбирали для идентификации клеточных линий, экспрессирующих hANGPTL4-специфичные антитела с применением анализов, описанных ниже. Анализы позволяли идентифицировать несколько клеточных линий, которые выделяли химерные антитела против hANGPTL4, которые обозначали как H1M222, H1M223, H1M224, H1M225, H1M234 и H1M236.
ANGPTL4-специфичные антитела человека также напрямую изолировали из антиген-иммунизированных B-клеток без слияния с миеломными клетками, как описано в U.S. 2007/0280945 A1. Вариабельные области тяжелой и легкой цепи клонировали с получением полностью антител человека против hANGPTL4, которые обозначали как H1H257, H1H268, H1H283, H1H284, H1H285, H1H291, H1H292, H1H295, H1H624, H1H637, H1H638, H1H644 и H1H653. Получали стабильные рекомбинантные антитело-экспрессирующие клеточные линии CHO.
Пример 2. Анализ применения вариабельного гена.
Для изучения структуры полученных антител клонировали и секвенировали нуклеиновые кислоты, кодирующие вариабельные области антитела. На основе последовательности нуклеиновой кислоты и предсказанной аминокислотной последовательности антитела определяли применение гена для каждой вариабельной области тяжелой цепи (HCVR) и вариабельной области легкой цепи (LCVR). В таблице 1 показано применение гена для выбранных антител согласно изобретению.
В таблице 2 показаны пары аминокислотных последовательностей вариабельных областей тяжелой и легкой цепи выбранных антител к hANGPTL4 и их соответствующие идентификаторы антител. Обозначения N, P и L относятся к антителам, содержащим тяжелые и легкие цепи с идентичными последовательностями CDR, но с вариациями последовательностей в областях, лежащим вне последовательностей CDR (т.е., в каркасных областях). Таким образом, варианты N, P и L определенного антитела содержат идентичные последовательности CDR в вариабельных областях тяжелой и легкой цепи, но содержат модификации в каркасных областях.
Пример 3. Определение аффинности связывания hANGPTL4.
Равновесные константы диссоциации (значения KD) для связывания антигена с выбранными антителами, при котором связываются аминокислотные остатки 26-148 ANGPTL4 человека, линейно слитого с IgG2a мыши (hANGPTL4-mFc; SEQ ID NO:480), определяли на основе поверхностной кинетики с применением биосенсорного поверхностного плазмонного резонансного анализа в реальном времени (BIACORE™ T100). hANGPTL4-mFc захватывали с применением поликлонального антитела против мыши IgG козы (GE Healthcare), которое химически соединяли с чипом BIACORE™ через свободные аминогруппы. Варьирующие концентрации (в диапазоне от 12,5 нМ до 50 нМ) антител к ANGPTL4 инъецировали через поверхность захваченного антигена в течение 90 секунд. Связывание антиген-антитело и диссоциацию отслеживали в реальном времени при 25°C и 37°C. Кинетический анализ проводили для определения KD и времени полужизни комплекса антиген/антитело диссоциация. Результаты показаны в таблице 3. Антитело человека против EGFR применяли в качестве отрицательного контроля, которое не демонстрировало связывания с захваченным hANGPTL4-mFc.
Для H1H268P и H1H284P фрагменты Fab получали посредством расщепления папаином и очистки стандартными способами очистки, и аффинность их связывания с hANGPTL4 измеряли при 25°C при pH 7,2 и pH 5,75 с применением системы BIACORE™, по существу согласно способу, описанному выше. В кратком изложении, различные концентрации (3,125 нМ-100 нМ) антител против hANGPTL4 (т.е., H1H268 Fab, полное H1H268 mAb, H1H284 Fab и полное H1H284 mAb) инъецировали через поверхность анти-mFc низкой плоности, захваченный hANGPTL4(26-148)-mFc (-68±4 РЕ), или поверхность аминосвязанного hANGPTL4(26-406)-His (R&D Systems) (450 РЕ) или аминосвязанной N-концевой области яванского макака (аминокислотные остатки 1-130 SEQ ID NO:490), экспрессированного с C-концевой гексагистидиновой меткой (MfANGPTL4(1-130)-His) (1,028 РЕ). Кинетический анализ проводили для измерения ka и kd, и рассчитывали значения KD и время полужизни комплекса антиген/антитело. Результаты показаны в таблице 4 (H1H268P) и таблице 5 (H1H284P).
Оба фрагмента Fab показали способность к связыванию со всеми формами ANGPTL4, хотя и с более низкой аффинностью, чем полные молекулы антитела.
Пример 4. Определение перекрестной реактивности анти-hANGPTL4 антител.
Возможную перекрестную реактивность антител к hANGPTL4 по отношению к родственным белкам, т.е., к hANGPTL3, ангиопоэтин-подобному белку (hANGPTL5) человека 5 и ANGPTL4 (mANGPTL4) мыши, тестировали для выбранных антител, т.е., H1H268P и H1H284P, с применением системы BIACORE™. В кратком изложении, антитела к hANGPTL4, а также отрицательные контроли, т.е., два моноклональных антитела (Контроль a и Контроль b), которые не связываются ни с одним из белков ANGPTL, инъецировали в концентрации 3,125 мкг/мл-50 мкг/мл через поверхности чипов аминосвязанного hANGPTL3-His (R&D Systems, каталожный № 3829-AN) при 5228 RU, hANGPTL4-His (R&D Systems, каталожный № 4487-AN) при 6247 RU, hANGPTL5-His (Abnova Corp., каталожный № H00253935-P01) при 5265 RU и mANGPTL4-His [R26-S410 mANGPTL4 (SEQ ID NO:478), слитого AS-линкером с C-концевой 6-гистидиновой меткой] при 5233 RU, соответственно. Также тестировали поликлональное антитело, специфичное к hANGPTL3 (R&D System, каталожный № BAF3485). Определяли связывание каждого антитела, выраженное в специальных единицах RU; результаты показаны в таблице 6.
H1H268P и H1H284P специфично связывались только с hANGPTL4-His и не связывались ни с одним другим родственным ANGPTL белком.
Кроме того, аффинность связывания H1H268P и H1H284P с различными пептидами ANGPTL3 и ANGPTL4 также определяли с использованием системы BIACORE™. В кратком изложении, H1H268P (1348±11 RU) и H1H284P (868±13 RU) захватывали через поверхность Fc против человека, и инъецировали различные концентрации (62,5 нМ-500 нМ) пептидов hANGPTL3 и hANGPTL4. Изучаемые пептиды представляли собой hANGPTL4 (R34-L66 SEQ ID NO:476), биотинилированный в N-концевой области hANGPTL4 (R34-L66 SEQ ID NO:476), hANGPTL3 (R36-I68 SEQ ID NO:485) и биотинилированный в N-концевой области hANGPTL3 (R36-I68 SEQ ID NO:485). Кинетический анализ проводили для измерения ka и kd, и рассчитывали значения KD и время полужизни комплекса антиген/антитело. Результаты показаны в таблице 7. NB: Связывание в экспериментальных условиях не описано.
| Таблица 7 | |||||
| Анти-hANGPTL4 антитело | Пептид | ks (M-1с-1) | kd (с-1) | KD (нМ) | t1/2 (мин) |
| H1H268P | hANGPTL3-Nконц. биотин | NB | - | - | - |
| hANGPTL4-Nконц. биотин | 4,53×103 | 2,94×10-4 | 64,8 | 39 | |
| hANGPTL3 | NB | - | - | - | |
| hANGPTL4 | 6,49×103 | 3,65×10-4 | 56,3 | 32 | |
Ни одно антитело не связывалось с пептидами hANGPTL3. Кроме того, H1H284P не связывалось ни с одним из пептидов hANGPTL4 даже при самой высокой применяемой концентрация пептида (500 нМ), в то время как H1H268P оказалось способным связываться с обоими пептидами hANGPTL4. Это позволяет предположить, что H1H268P распознает линейный эпитоп внутри области 34-66. Напротив, H1H284P либо связывается с участком, лежащим за пределами этой области, либо не распознает линейный эпитоп в этой области.
Пример 5. Ингибирование hANGPTL4 анти-ANGPTL4 антителами.
Липопротеинлипаза (LPL) играет критическую роль в метаболизме липидов у человека. LPL катализирует гидролиз триглицеридов и инициирует метаболизирование жирных кислот. ANGPTL4 ингибирует активность LPL, что приводит к повышению уровня липидов (Oike et al., 2005, Trends in Molecular Medicine 11(10):473-479). N-концевая суперспиральная область ANGPTL4 претерпевает гомомультимеризацию, как изолированно, так и при присоединении к C-концевой фибриноген-подобной области. N-концевая область также ингибирует LPL при экспрессии без C-концевой фибриногенной области. Разработаны бесклеточные биотесты для определения способности выбранных антител к hANGPTL4 ингибировать индуцированное ANGPTL4 снижение активности LPL.
Ингибирование активности hANGPTL4 выбранными антителами к hANGPTL4 определяли с применением непрерывного флюорометрического теста на липазу CONFLUOLIP™ (Progen, Germany) с применением двух белков hANGPTL4: полноразмерного hANGPTL4 (т.е., аминокислотные остатки 26-406 SEQ ID NO:476) с C-концевой гексагистидиновой меткой (hANGPTL4-His; R&D Systems, MN) и hANGPTL4-mFc (SEQ ID NO: 480), содержащим N-концевой суперспиральный участок.
В кратком изложении, 2 нМ сывороточного LPL, 0,25 мкΜ ApoCII человек (кофактор LPL), 2 мг/мл BSA и 1,6 мМ CaCl2 предварительно смешивали в 96-луночном планшете. К смеси Apo/LPL добавляли белок hANGPTL4-His или hANGPTL4-mFc до конечной концентрации, составляющей 10 нМ и 2 нМ, соответственно. Смеси белков Apo/LPL/ANGPTL4 затем добавляли к серийно разведенным антителам к hANGPTL4 с начальной концентрацией, составляющей 300 нМ (для ингибирования hANGPTL4-His) или 100 нМ (для ингибирования hANGPTL4-mFc) и инкубировали при комнатной температуре в течение 30 минут (конечный объем 50 мкл). После инкубации к смеси антител добавляли 200 мкл восстановленного липазного субстрата, 1-тринитрофенил-амино-додеканоил-2-пирендеканоил-3-0-гексадецил-sn-глицерина (LS-A, Progen) и инкубировали при 37°C в течение двух часов. Затем измеряли флуоресценцию при 342 нм/400 нм (возбуждение/испускание) с использованием микроспектрофотометра для чтения планшетов FlexStation® 3 (Molecular Devices, CA). Флуоресценция прямо пропорциональна активности LPL. Результаты показаны в таблице 8. Контроль I: поликлональное антитело кролика, специфичное к hANGPTL4 (BioVendor). Контроль II: неподходящее антитело человека, которое не связывает hANGPTL.4. NT: не изучено. Суммарное ингибирование (т.е., 100% ингибирование) определяли на основе относительных флуоресцентных единиц (ОФУ) с 2 нМ сывороточной LPL, 0,25 мкΜ ApoCII человека, 2 мг/мл BSA и 1,6 мМ CaCl2, в отсутствие антител против ANGPTL4 и антител ANGPTL4.
| Таблица 8 | ||
| Антитело | % ингибирования активности hANGPTL4 | |
| hANGPTL4-mFc (2 нМ) |
hANGPTL4-His (10 нМ) |
|
| H1H236N2 | 13 | 19 |
| H1H257P | 16 | NT |
| H1H268P | 76 | 80 |
| H1H284P | 82 | 90 |
| H1H285P | 28 | 47 |
| H1H292P | 20 | 53 |
| H1H624P | 62 | 48 |
| H1H653P | 55 | 48 |
| Контроль I | 29 | 66 |
| Контроль II | Отсутствие ингибирования | Отсутствие ингибирования |
Антитела H1H284P и H1H268P среди изученных антител продемонстрировали самое высокое подавление ингибирующей активности ANGPTL4 против LPL, включая контрольное поликлональное антитело hANGPTL4. Для антител H1H284P и H1H268P концентрации антител, необходимые для 50% максимального ингибирования (IC50) 2 нМ hANGPTL4-mFc определяли как 0,8 нМ и 1,2 нМ, соответственно. Кроме того, концентрации антител, необходимые для 50% максимального ингибирования 10 нМ hANGPTL4-His, определяли как 0,5 нМ и 0,2 нМ, соответственно.
Так же, H1H284P и H1H268P тестировали в биотесте LPL на их способность ингибировать межвидовые ортологи: N-концевую область (аминокислотные остатки 26-148) яванского макака, экспрессированную с N-концевой гексагистидиновой меткой (His-MfANGPTL4; SEQ ID NO:488) и полноразмерный ортолог мыши (аминокислотные остатки 26-410 SEQ ID NO:478) с C-концевой гексагистидиновой меткой (mANGPTL4-His). Сначала был получен полный дозозависимый эффект с применением белка ANGPTL4 в анализе LPL для определения EC50 ANGPTL4 для каждого эксперимента, а затем определение IC50 для каждого антитела проводили с применением постоянной концентрации белка ANGPTL4, как показано в таблице 9. Концентрации антител варьировали от 0 до 100 нМ. NB: Отсутствие блокирования.
| Таблица 9 | |||||
| hANGPTL4(26-406)-His | hANGPTL4 (26-148)-mFc |
His-MfANGPTL4 (26-148) |
mANGPTL4 (26-148)-His |
||
| EC50 (нМ) | 6,00 | 0,50 | 3,89 | 0,63 | |
| Постоянный ANGPTL4 (нМ) | 10 | 2 | 10 | 1 | |
| EC50 (нМ) | H1H268P | 0,46 | 0,47 | 0,42 | NB |
| H1H264P | 0,31 | 0,51 | 0,42 | NB | |
| IgG1 контроль | NB | NB | NB | NB | |
Оба антитела ингибировали активность как ANGPTL4 человека (полноразмерного и N-концевого), так и ANGPTL4 обезьяны (N-концевого) с IC50, составляющими приблизительно 0,3-0,5 нМ; но ни одно антитело не ингибировало ANGPTL4 мыши (полноразмерный) даже с самой высокой изучаемой концентрацией антитела (т.е., 100 нМ).
Пример 6. Воздействие ANGPTL4 in vivo на уровни липидов в плазме.
hANGPTL4 внутривенно вводили мышам C57BL/6 для определения биологического эффекта hANGPTL4 на уровни липидов в плазме. В кратком изложении, мышей C57BL/6 разделяли на четыре группы по пять животных, и каждой группе вводили различное количество белка hANGPTL4-mFc (SEQ ID NO:480): 25 мкг, 50 мкг, 100 мкг и 300 мкг на мышь. Контрольная группа получала инъекции PBS. Белок hANGPTL4-mFc и PBS вводили посредством внутривенной инъекции (в./в.) в хвостовую вену. Затем у мышей брали кровь через 15 мин, 30 мин, 60 мин и 120 мин после доставки hANGPTL4-mFc или PBS и определяли уровни липидов в плазме посредством химической системы ADVIA® 1650 (Siemens). Для каждой группы дозирования определяли уровни триглицеридов, общего холестерина, липопротеинов низкой плотности (LDL), неэтерифицированных жирных кислот (NEFA-C) и липопротеинов высокой плотности (HDL). Уровни общего холестерина, LDL, NEFA-C и HDL значительно не различались между группами дозирования на каждый момент времени после инъекции. Инъекции 25 мкг/мышь hANGPTL4-mFc повышали уровень циркулирующих триглицеридов более чем в два раза по сравнению с контрольными мышами (PBS) через 30 минут после инъекции. Таким образом, дозу в 25 мкг hANGPTL4-mFc выбирали в качестве возможной минимальной дозы для анализа ингибирования ANGPTL4-индуцированного повышения сывороточного уровня триглицеридов в результате воздействия выбранных антител к hANGPTL4, как описано ниже.
Пример 7. Ингибирование ANGPTL4 in vivo анти-ANGPTL4 антителами.
В другой серии экспериментов выбранные антитела к hANGPTL4 тестировали на их способность ингибировать hANGPTL4-индуцированное повышение уровня триглицеридов. Также измеряли уровни общего холестерина (Total-C), LDL, NEFA-C и HDL. В кратком изложении, мышей C57BL/6 разделяли на группы по пять мышей в каждой для каждого изучаемого антитела. Антитела вводили в дозе 5 мг/кг посредством подкожной инъекции. Контрольной группе I, т.е. мышам, которые не получали ни антител против hANGPTL4, ни hANGPTL4, вводили PBS. Через двадцать четыре часа после инъекции антител каждой группе, получавшей антитела, вводили (в./в.) hANGPTL4-mFc (SEQ ID NO:480) в дозе 25 мкг/мышь. У мышей брали кровь через 30 мин после инъекции hANGPTL4-mFc и определяли уровни липидов посредством химической системы ADVIA® 1650 (Siemens). Для каждого уровня триглицеридов, общего холестерина, LDL, NEFA-C и HDL для каждой группы, получавшей антитела, или контрольной группы, рассчитывали средние значения. Уровень циркулирующих антител к hANGPTL4 (сывороточный Ab) также определяли посредством стандартного анализа ELISA. В кратком изложении, планшеты покрывали антителами Fc козы против человека (Sigma-Aldrich) для захвата сывороточного Ab. Затем к планшетам добавляли сыворотку и определяли захваченные антитела к hANGPTL4 на основе хемилюминисценции с применением пероксидазы хрена (HRP), конъюгированной с антителами IgG козы против человека (Sigma-Aldrich). Результаты, выраженные в виде (средних значений ± SEM) сывороточных концентраций липидов, показаны в таблице 10-12. Контроль I: Мыши, получавшие PBS, но не получавшие ни антител к hANGPTL4, ни антител к hANGPTL4-mFc. Контроль II: Мыши, получавшие антитела человека, специфичные к CD20 (т.е., mAb с последовательностью клона 2F2, раскрытой в US 2008/0260641), и hANGPTL4-mFc.
| Таблица 10 | ||||||
| Антитело | Триглицериды (мг/дл) | Общий хол. (мг/дл) | LDL (мг/дл) | NEFA-С (мг/дл) | HDL (мг/дл) | Сывороточный Ab (мкг/мл) |
| Контроль I | 98,20±5,49 | 89,80±4,28 | 5,60±0,66 | 1,01±0,04 | 44,18±2,43 | - |
| Контроль II | 211,60±58,29 | 93,40±5,52 | 6,30±0,22 | 1,33±0,17 | 44,62±3,14 | 12,76±0,52 |
| H1H284P | 99,20±9,52 | 80,80±6,40 | 4,98±0,87 | 0,99±0,11 | 39,60±3,46 | 7,96±0,55 |
| H1H257P | 115,80±6,43 | 84,40±3,53 | 5,30±0,36 | 0,97±0,03 | 41,38±3,24 | 8,43±0,86 |
| Таблица 11 | ||||||
| Антитело | Триглицериды (мг/дл) | Общий хол. (мг/дл) | LDL (мг/дл) | NEFA-С (мг/дл) | HDL (мг/дл) | Сывороточный Ab (мкг/мл) |
| Контроль I | 66,60±7,94 | 70,00±2,3 | 3,88±0,36 | 0,76±0,08 | 35,26±1,09 | - |
| Контроль II | 161,00±17,83 | 73,60±0,93 | 4,12±0,17 | 1,18±0,09 | 35,10±0,6 | 11,05±2,28 |
| H1H236N2 | 151,80±9,26 | 72,40±1,81 | 4,26±0,25 | 1,11±0,118 | 33,78±1,13 | 9,20±0,63 |
| H1H624P | 81,20±9,26 | 72,80±5,49 | 4,36±0,92 | 0,86±0,07 | 35,40±2,68 | 11,76±0,89 |
| H1H268P | 92,60±11,44 | 76,00±2,14 | 4,94±0,51 | 0,82±0,04 | 35,94±1,64 | 8,05±1,06 |
После инъекции hANGPTL4-mFc (25 мкг) большая часть изучаемых антител к hANGPTL4 (см. таблице 10-12) демонстрировала значительное повышение сывороточного уровня триглицеридов по сравнению с мышами, обработанными неподходящими антителами (Контроль II).
Пример 8. Получение анти-ANGPTL4 антител с изотипом hIgG4.
Антитела H1H268P и H1H284P с изотипом hIgG1 трансформировали в антитела с изотипом hIgG4 посредством замены соответствующих константных областей на аминокислотную последовательность hIgG4 SEQ ID NO:483, содержащую мутацию S108P в шарнирной области. Кроме того, в каркасную область 1 H1H268P (SEQ ID NO:42) вносили единичную замену аминокислоты с получением H4H268P2 (SEQ ID NO:487) изотипа IgG4. KD(пМ) и значения t1/2 антител IgG4, обозначаемых как H4H268P2 и H4H284P, соответственно, для связывания hANGPTL4-mFc (SEQ ID NO:480) получали с использованием Biacore при pH 7,4 и 25°C, согласно протоколу, описанному в примере 3 выше. Результаты показаны в таблице 13 ниже.
| Таблица 13 | ||
| Антитело | KD (пМ) | t1/2 (мин) |
| H4H268P2 | 146 | 195 |
| H4H284P | 143 | 205 |
H4H268P2 и H4H284P и соответствующие версии IgG1, H1H268P и H1H284P, соответственно, тестировали в анализе ингибирования LPL, как описано в примере 5 выше, для определения значений IC50. Результаты показаны в таблице 14. NB: Отсутствие блокирования.
| Таблица 14 | |||
| hAngPTL4(26-406)-His | hANGPTL4(26-148)-mFc | ||
| EC50 (нМ) | 4,54 | 0,29 | |
| Константа ANGPTL4 (нМ) | 10 | 2 | |
| IC50 (нМ) | H1H268P | 0,20 | 0,67 |
| H1H284P | 0,42 | 0,33 | |
| Контроль IgG1 | NB | NB | |
| H4H268P2 | 1,61 | 1,85 | |
| H4H284P | 1,19 | 1,69 | |
| Контроль IgG4 | NB | NB | |
В этом анализе H1H268P и H1H284P продемонстрировали IC50 в диапазоне приблизительно 0,2-0,7 нМ для полноразмерного и N-концевого белков hANGPTL4, в то время как H4H268P2 и H4H284P продемонстрировали IC50 в диапазоне приблизительно от 1,0 до 2,0 нМ.
Пример 9. Анализ фармакокинетики анти-ANGPTL4 антител.
Определяли фармакокинетические скорости выведения анти-hANGPTL4 антител H4H268P2 и H4H284P у мышей дикого типа и у трансгенных мышей, экспрессирующих ANGPTL4 человека [hANGPTL4(+/+) мыши]. Штаммы для мышей дикого типа и трансгенных мышей представляли собой C57BL6 (75%) и 129Sv (25%). Отдельные группы, состоящие из 5 мышей дикого типа или мышей hANGPTL4(+/+), получали подкожно 1 мг/кг H4H268P2, H4H284P или соответствующего изотипу (hIgG4) контроля с неподходящей специфичностью. Образцы крови собирали через 0 часов, 6 часов, 1 сутки, 2 суток, 3 суток, 4 суток, 7 суток, 10 суток, 15 суток, 22 суток и через 30 суток после инъекции. Сывороточные уровни антител человека определяли посредством ELISA «сэндвич»-типа. В кратком изложении, поликлональные антитела козы против IgG человека (Fc-специфичные) (Jackson ImmunoResearch, PA) вносили в 96-луночные планшеты в концентрации, составляющей 1 мкг/мл, и инкубировали в течение ночи при 4°C. Затем планшеты блокировали BSA, в планшеты добавляли образцы сыворотки в шестикратном серийном разведении и стандартные образцы соответствующих антител в двенадцатикратном серийном разведении и инкубировали в течение одного часа при комнатной температуре. После отмывания подходящим отмывочным буфером захваченные антитела человека обнаруживали с применением тех же поликлональных антител козы против IgG человека (Fc-специфических), конъюгированных с пероксидазой хрена (HRP) (Jackson ImmunoResearch, PA), и детектировали стандартный колориметрический ответ с использованием тетраметилбензидина (TMB), измеряя оптическую плотность при 450 нм в спектрофотометре для чтения планшетов. Концентрации антител человека в сыворотке определяли с применением стандартной кривой, полученной для того же планшета с образцами. Результаты показаны в таблице 15 и фиг. 3A и 3B.
| Таблица 15 | |||
| Антитело | Генотип мыши | Cmax (мкг/мл) | AUC (ч*мкг/мл) |
| H4H268P2 | Дикий тип | 18,4 | 318 |
| H4H284P | Дикий тип | 15,7 | 200 |
| Контроль hIgG4 | Дикий тип | 14,2 | 199 |
| H4H268P2 | hANGPTL4(+/+) | 13,3 | 37,0 |
| H4H284P | hANGPTL4(+/+) | 5,86 | 7,60 |
| Контроль hIgG4 | hANGPTL4(+/+) | 11,6 | 168 |
Как показано в таблице 15, оба антитела к hANGPTL4 демонстрировали такие же скорости выведения, как и соответствующее изотипу контрольное антитело у мышей дикого типа, что видно из области под кривой (AUC), составляя через 30 суток приблизительно 318, 200 и 199 (час*мкг/мл), соответственно, для H4H268P2, H4H284P и контроля hIgG4 (также см. фиг. 3A). У трансгенных мышей, экспрессирующих только ANGPTL4 [hANGPTL4(+/+)] человека, скорости выведения, как видно из AUC, были выше как для H4H268P2 (37,0 час*мкг/мл), так и для H4H284P (7,60 час*мкг/мл) по сравнению со скоростями выведения у мышей дикого типа (318 и 200 час*мкг/мл, соответственно) и по сравнению со скоростью выведения соответствующего изотипу контрольного антитела как у мышей hANGPTL4(+/+) (168 час*мкг/мл), так и у мышей дикого типа (199 час*мкг/мл) (также см. фиг. 3B). У мышей hANGPTL4(+/+) AUC через 30 суток для H4H284P (7,60 час*мкг/мл) был приблизительно в 5 раз меньше, чем AUC для H4H268P2 (37,0 час*мкг/мл). Эти результаты позволяют предположить, что оба антитела к hANGPTL4 демонстрируют мишень-опосредованный клиренс у мышей, экспрессирующих ANGPTL4 человека, и H4H284P демонстрирует значительно более высокую скорость выведения, чем H4H268P2.
Пример 10. Эффект in vivo анти-hANGPTL4 антител IgG1 на циркулирующий уровень TG у гуманизированных ANGPTL4 мышей.
Эффект антител к hANGPTL4 H1H268P и H1H284P на сывороточный уровень TG определяли у мышей, экспрессирующих белок ANGPTL4, содержащий N-концевую биспиральную область человека ("гуманизированные ANGPTL4 мыши"). Гуманизированных ANGPTL4 мышей получали посредством замены первых трех экзонов гена мыши Angptl4 (N-концевой биспиральный участок) на соответствующую N-концевую биспиральную последовательность ANGPLT4 человека в эмбриональных стволовых клетках C57BL6/129 (F1H4). После установления трансмиссии зародышевой линии гетерозиготных мышей (ANGPTL4hum/+) скрещивали с получением гомозиготных мышей [ANGPTL4hum/hum или hANGPTL4(+/+)] на основе C57BL6. У гуманизированных ANGPTL4 мышей брали кровь за 7 суток до эксперимента (сутки -7) и разделяли на группы по шесть мышей в каждой для каждого изучаемого антитела. Антитела (H1H268P, H1H284P и соответствующее изотипу (hIgG1) контрольное антитело, не проявляющее перекрестной реактивности по отношению к антигенам мыши) вводили в дозе 10 мг/кг посредством подкожной инъекции.
После 4-часового голодания у мышей брали кровь через 1, 4, 7 и 11 суток после инъекции антител; и определяли сывороточные уровни TG посредством химической системы ADVIA® 1800 (Siemens). Для каждого момента времени и для каждого антитела средние уровни TG рассчитывали. Результаты, выраженные в виде (средних значений ± SEM) сывороточных концентраций TG, показаны в таблице 16.
| Таблица 16 | |||
| Сутки после инъекции | Сывороточный TG (мг/дл) | ||
| hIgG1 Контроль | H1H268P | H1H284P | |
| -7 | 117±18 | 112±9,3 | 113±11 |
| 1 | 138±21 | 129,8±5,6 | 125±18 |
| 4 | 102±14 | 73,3±8,9 | 67±9,8 |
| 7 | 112±10 | 83±14 | 91±7,2 |
| 11 | 110±15 | 76±5,5 | 109±11 |
Уровень циркулирующих антител к hANGPTL4 ("сывороточные Ab человека") также определяли посредством стандартного анализа ELISA. В кратком изложении, планшеты покрывали антителами козы против Fc человека (Sigma-Aldrich) для захвата сывороточных Ab человека. Затем в планшеты добавляли сыворотку и обнаруживали захваченные антитела к hANGPTL4 посредством хемилюминисценции с применением конъюгированных с пероксидазой хрена (HRP) антител козы против IgG человека (Sigma-Aldrich). Результаты, выраженные в виде (средних значений ± SEM) сывороточных Ab человека, показаны в таблице 17.
| Таблица 17 | |||
| Сутки после инъекции | Сывороточные антитела человека (мкг/мл) | ||
| hIgG1 Контроль | H1H268P | H1H284P | |
| -7 | 2,71±2,18 | 2,92±2,93 | 3,02±2,38 |
| 1 | 24384±911 | 24130±1788 | 16459±1455 |
| 4 | 22553±1811 | 16557±1369 | 9103±767 |
| 7 | 13833±467 | 12586±1176 | 2428±525 |
| 11 | 13145±1598 | 6106±1111 | 135±38 |
Введение H1H268P гуманизированным ANGPTL4 мышам приводило к ~25-30% уменьшению уровня циркулирующих TG через 4-11 суток после введения антител по сравнению с мышами, которым вводили соответствующее изотипу контрольное антитело. Снижение уровня TG, вызванное введением H1H284P, было самым значительным через 4 суток после инъекции антитела (сокращение TG на ~34%), но через 11 суток уровень TG снова повышался до контрольного уровня, возможно, из-за высокой скорости выведения антител.
Пример 11. Эффект in vivo анти-hANGPTL4 антител IgG4 на уровень циркулирующих TG у гуманизированных ANGPTL4 мышей.
Определяли эффект антител к hANGPTL4, H4H268P2 и H4H284P на сывороточный уровень TG у гуманизированных ANGPTL4 мышей. У гуманизированных ANGPTL4 мышей брали кровь за 7 суток до эксперимента, и мышей разделяли на группы по шесть в каждой для каждого изучаемого антитела. Антитела (H4H268P2, H4H284P и соответствующий изотипу (hIgG4) контроль, не проявляющий перекрестной реактивности по отношению к антигенам мыши) вводили в дозе 10 мг/кг посредством подкожной инъекции. У мышей брали кровь после 4-часового голодания через 1, 4, 7 и 11 суток после инъекции антител; и определяли уровни TG в сыворотке с применением химической системы ADVIA® 1800 (Siemens). Рассчитывали средние уровни TG для каждого момента времени и для каждого антитела. Результаты, выраженные в виде (средних значений ± SEM) сывороточных концентраций TG, показаны в таблице 18.
| Таблица 18 | |||
| Сутки после инъекции | Сывороточный TG (мг/дл) | ||
| hIgG4 Контроль | H4H268P2 | H4H284P | |
| -7 | 103±9,5 | 101±9,3 | 103±8,0 |
| 1 | 118±13 | 81±6,8 | 86±8,6 |
| 4 | 115±9,8 | 67±5,3 | 69±6,4 |
| 7 | 81±9,7 | 56±7,1 | 71±11 |
| 11 | 109±10 | 87±8,7 | 83±7,3 |
Введение H4H268P2 и H4H284P гуманизированным ANGPTL4 мышам приводило к значительному снижению уровней циркулирующих TG через 1 сутки (H4H268P2) и через 4 суток (H4H268P2 и H4H284P) после введения антител по сравнению с мышами, которым вводили соответствующее изотипу контрольное антитело.
Эффект H4H268P2 на уровни циркулирующих TG далее изучали у гуманизированных ANGPTL4 мышей, скрещенных с нулевыми по ApoE мышами. Модель нулевых по ApoE мышей известна как высоко атерогенная и гиперлипидемическая модель, в которой большая часть холестерина и TG циркулирует в виде частиц VLDL в результате нарушенного остаточного клиренса VLDL. У гибридов гуманизированных ANGPTL4 и нулевых по ApoE мышей брали кровь за 7 суток до эксперимента, и мышей разделяли на 2 группы по шесть мыши в каждой. Антитела H4H268P2 и контрольные Ab вводили в дозе 10 мг/кг посредством подкожной инъекции. У мышей брали кровь после 4-часового голодания через 1, 4, 7, 11 и 17 суток после инъекции антител и определяли уровни TG в сыворотке с применением химической системы ADVIA® 1800 (Siemens). Снижения уровня TG, показанные на фиг. 4, выражены в процентах от уровней TG по сравнению с контрольными Ab.
Уровни TG значительно сокращались в течение всех 17 суток (более чем на 42%) с самым заметным сокращением через 7 суток (~50%) после введения H4H268P2 по сравнению с мышами, обработанными контрольными Ab.
Пример 12. Эффект in vivo анти-hANGPTL4 антител в комбинации с фенофибратом на сывороточный уровень TG.
Оценивали эффекты антител к ANGPTL4 H4H268P2 и сокращающего уровень TG лекарственного средства фенофибрат, как по отдельности, так и в комбинации, на сывороточные уровни TG у гуманизированных ANGPTL4 мышей. У мышей брали кровь за 7 суток до эксперимента после 4-часового голодания, и мышей разделяли на 4 группы по шесть мышей в каждой. Группам 2 и 4 вводили H4H268P2 в дозе 10 мг/кг посредством подкожной инъекции на сутки 0, и группу 1 (контрольная группа), а группу 2 держали на обычной пище. Группы 3 и 4 получали пищу, обогащенную 0,05% (масс./масс.) фенофибрата (режимы дозирования определяли экспериментально в пилотном исследовании). Сыворотку собирали через 7 суток после введения H4H268P2 и/или фенофибрата (после 4-часового голодания) и анализировали с применением химической системы ADVIA® 1800 (Siemens). Результаты показаны на фиг. 5.
Введение H4H268P2 отдельно и в комбинации с фенофибратом приводило к значительному сокращению уровня циркулирующих TG через 7 суток после введения. Уровни TG сокращались на ~40% (в среднем) через 7 суток после введения H4H268P2 отдельно, на ~25% после введения фенофибрата отдельно и на ~50% после введения комбинации H4H268P2 и фенофибрата по сравнению с контрольной группой, получавшей обычную пищу. H4H268P2 продемонстрировал большую эффективность, чем фенофибрат, в сокращении уровня циркулирующих TG на мышиной модели. Комбинированная обработка продемонстрировала синергическое воздействие H4H268P2 и фенофибрата на уровень TG. Стоит заметить, что печень, которую брали у мышей, получавших фенофибрат (группы 3 и 4) в течение 7 суток, была сильно увеличенной (в 1,8 раз, масса печени/масса тела) по сравнению с мышами, получавших контрольную пищу (группы 1 и 2) (данные не показаны).
Пример 13. Пилотное исследование фармакокинетики/фармакодинамики анти-ANGPTL4 антител у страдающих ожирением макак-резусов.
Этап I: В пилотном исследовании фармакокинетики/фармакодинамики (PK/PD), не соответствующем GLP, страдающим ожирением макакам-резусам (Macaca mulatta) H4H268P2 и H4H284P вводили болюсной внутривенной (в/в) инъекцией. Макак-резусов выбирали потому, что этот вид является близкородственным человеку, как филогенетически, так и физиологически, и представляет собой вид, который широко используют для неклинических оценок токсичности. Страдающих ожирением обезьян, которые получали пищу с высоким содержанием жиров в течение более 6 месяцев, выбирали потому, что такие животные, как правило, демонстрируют умеренно повышенные уровни TG (т.е., в среднем >100 мг/дл; гипер-TG). Восемь здоровых самцов обезьян акклиматизировали и назначали к исследованию с оценкой исходного состояния в течение 7 суток перед началом лечения. Все животные получали носитель (10 мМ гистидин, pH 6) посредством внутривенного вливания на сутки -5, после чего четыре из них получали H4H268P2, а другие четыре получали H4H284P в дозе 10 мг/кг в/в на сутки 0. Никаких реакций в месте инъекции или других неблагоприятных воздействий не наблюдали ни в один момент времени после вливания.
Образцы сыворотки собирали начиная с исходного периода по сутки 35 после лечения и устанавливали сывороточные уровни липидов с использованием химической системы ADVIA® 1800 (Siemens). Средние исходные значения для каждого животного определяли на основе образцов, взятых на сутки -7, -5 и 0. Предварительный анализ образцов, взятых после ведения носителя, показал, что 3 страдающих ожирением животных из каждой группы демонстрировали повышенные уровни TG после голодания (т.е., TG > 100 мг/дл), причем одно животное из каждой группы демонстрировало уровень TG, находящийся в пределах нормального диапазона (т.е., среднее значение уровня TG после голодания, составляющее 42 мг/дл и 84 мг/дл). Таким образом, анализ результатов проводили для 3 животных в каждой группе. Для каждого животного определяли процент (%) изменений сывороточных уровней TG по сравнению с исходным значением и усредняли значения для каждой группы Ab. Результаты показаны на фиг. 6.
Введение H4H268P2 трем умеренно гипер-TG животным приводило к максимальному сокращению на 57% через 4 суток после лечения. Средние сывороточные уровни TG для этих животных после лечения H4H268P2 оставались равными или были ниже 100 мг/дл вплоть до приблизительно 25 суток. Умеренные эффекты наблюдали для дополнительных липидов, таких как LDL-холестерин (LDL-C) и HDL-C; однако уровень общего холестерина не изменялся (данные не показаны). Введение H4H268P2 одному страдающему ожирению животному с низкими уровнями TG не показало никакого достоверного эффекта на снижение уровней TG после голодания или другие показатели уровней липидов (данные не показаны), что позволяет предположить, что для антитела существует более низкий предел сокращения уровней TG.
Этап II: В пилотном исследовании фармакокинетики/фармакодинамики (PK/PD), не соответствующем GLP, восьми страдающим ожирением макакам-резусам (Macaca mulatta) H4H268P2 вводили болюсной внутривенной (в/в) инъекцией. Этап введения носителя в этом исследовании пропускали. Три оценки исходного состояния проводили на сутки -7, -3 и 0 исследования, и все восемь обезьян получали H4H268P2 в дозе 10 мг/кг посредством в/в на сутки 0. Образцы сыворотки собирали начиная с исходного периода по сутки 35 после лечения и устанавливали сывороточные уровни липидов с применением химической системы ADVIA® 1800 (Siemens). Животных разделяли на группы для анализа результатов на основе средних исходных уровней TG с тем, чтобы предсказать эффект: A. TG < 150 мг/дл (n=3); B. 150 мг/дл < TG < 500 мг/дл (n=4); и C. TG > 1000 мг/дл (n=1).
На фиг. 7 представлены уровни TG для трех групп, выраженные в виде процента изменений уровней TG по сравнению с 3 исходными уровнями TG. Как и ожидалось на основе доклинических данных, самое значительное сокращение уровней TG после голодания наблюдалось у животных с более высокими исходными сывороточными уровнями TG. Особенно значительное и быстрое сокращение уровня TG наблюдали у животного, у которого уровень TG составлял > 1000 мг/дл. Значительное сокращение уровней TG (50-68%) наблюдали у животных с исходными уровнями 150 мг/дл < TG < 500 мг/дл. В этой группе страдающих ожирением обезьян введение H4H268P2 повышало HDL-C, но не влияло на LDL-C или общий холестерин (данные не показаны). Животные с исходным уровнем TG < 150 мг/дл (нормальные уровни TG) не отвечали на лечение H4H268P2.
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Regeneron Pharmaceuticals, Inc.
<120> АНТИТЕЛА ЧЕЛОВЕКА ПРОТИВ АНГИОПОЭТИН-ПОДОБНОГО БЕЛКА 4 ЧЕЛОВЕКА
<130> 6250A-WO
<140> To be assigned
<141> Filed herewith
<150> 61/290,092
<151> 2009-12-24
<150> 61/306,359
<151> 2010-02-19
<150> 61/328,316
<151> 2010-04-27
<150> 61/349,273
<151> 2010-05-28
<150> 61/356,126
<151> 2010-06-18
<160> 492
<170> FastSEQ for Windows Version 4.0
<210> 1
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1
caggtgcagc tggtgcagtc tgggggaggc ttggtgcagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cactttcagt agctacgaca tgcactgggt ccgccaagtt 120
gcaggaaaag gtctggagtg ggtctcagcc attggtactg ctggtgacac atactatcca 180
gtctccgtga agggccgatt caccatctct agagaaaatg ccaagaactc cttgtatctt 240
cacatgaaca gcctgagagc cggggacacg gctgtgtatt tctgtgcaag aggagacagt 300
agaaactact acgttgggga ctactttgac tactggggcc agggaaccac ggtcaccgtc 360
tcctca 366
<210> 2
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 2
Gln Val Gln Leu Val Gln Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Val Ala Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Pro Val Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
His Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Phe Cys Ala
85 90 95
Arg Gly Asp Ser Arg Asn Tyr Tyr Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 3
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 3
ggattcactt tcagtagcta cgac 24
<210> 4
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 4
Gly Phe Thr Phe Ser Ser Tyr Asp
1 5
<210> 5
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 5
attggtactg ctggtgacac a 21
<210> 6
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 6
Ile Gly Thr Ala Gly Asp Thr
1 5
<210> 7
<211> 48
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 7
gcaagaggag acagtagaaa ctactacgtt ggggactact ttgactac 48
<210> 8
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 8
Ala Arg Gly Asp Ser Arg Asn Tyr Tyr Val Gly Asp Tyr Phe Asp Tyr
1 5 10 15
<210> 9
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 9
gccatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt aggtggttgg cctggtatca gcaaaaacca 120
gggaaagccc ctaaggtcct gatctatcag gcgtccaatt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag tatgatagtt attctcgggc gttcggccga 300
gggaccaagg tggaaatcaa a 321
<210> 10
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 10
Ala Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Arg Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Tyr Gln Ala Ser Asn Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Ser Tyr Ser Arg
85 90 95
Ala Phe Gly Arg Gly Thr Lys Val Glu Ile Lys
100 105
<210> 11
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 11
cagagtatta gtaggtgg 18
<210> 12
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 12
Gln Ser Ile Ser Arg Trp
1 5
<210> 13
<211> 9
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 13
caggcgtcc 9
<210> 14
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 14
Gln Ala Ser
1
<210> 15
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 15
caacagtatg atagttattc tcgggcg 27
<210> 16
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 16
Gln Gln Tyr Asp Ser Tyr Ser Arg Ala
1 5
<210> 17
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 17
gaggtgcagc tggtggagtc tgggggaggc ttggtgcagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cactttcagt agctacgaca tgcactgggt ccgccaagtt 120
gcaggaaaag gtctggagtg ggtctcagcc attggtactg ctggtgacac atactatcca 180
gtctccgtga agggccgatt caccatctct agagaaaatg ccaagaactc cttgtatctt 240
cacatgaaca gcctgagagc cggggacacg gctgtgtatt tctgtgcaag aggagacagt 300
agaaactact acgttgggga ctactttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 18
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 18
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Val Ala Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Pro Val Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
His Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Phe Cys Ala
85 90 95
Arg Gly Asp Ser Arg Asn Tyr Tyr Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 19
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 19
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt aggtggttgg cctggtatca gcaaaaacca 120
gggaaagccc ctaaggtcct gatctatcag gcgtccaatt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag tatgatagtt attctcgggc gttcggccga 300
gggaccaagg tggaaatcaa a 321
<210> 20
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 20
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Arg Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Tyr Gln Ala Ser Asn Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Ser Tyr Ser Arg
85 90 95
Ala Phe Gly Arg Gly Thr Lys Val Glu Ile Lys
100 105
<210> 21
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 21
gaggtgcagc tggtggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cactttcagt agctacgaca tgcactgggt ccgccaagct 120
acaggaaaag gtctggagtg ggtctcagct attggtactg ctggtgacac atactatcca 180
ggctccgtga agggccgatt caccatctcc agagaaaatg ccaagaactc cttgtatctt 240
caaatgaaca gcctgagagc cggggacacg gctgtgtatt actgtgcaag aggagacagt 300
agaaactact acgttgggga ctactttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 22
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 22
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Ala Thr Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Pro Gly Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Arg Asn Tyr Tyr Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 23
<211> 322
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 23
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt aggtggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatcag gcgtccagtt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag tatgatagtt attctcgggc gttcggccaa 300
gggaccaagg tggaaatcaa ac 322
<210> 24
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 24
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Arg Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gln Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Ser Tyr Ser Arg
85 90 95
Ala Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 25
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 25
caggtacagc tgcagcagtg gggcgcagga ctgttgaagc cttcggagac cctgtccctc 60
acctgcactg tctatggtgg atccttcagt attcatcact ggacctggat ccgccatccc 120
ccagggaagg ggctggagtg gattggggag atcaatcatc gtggaagcac caactacaac 180
ccgtccctca agagtcgagt caccatatca atagacacgt ccaagaacca gttctccctg 240
aagctgagcg ctgtgaccgc cgcggacacg gctgtatatt actgtgcgag aggcttacga 300
tttttggact ggttatcgtc ctactttgac tactggggcc agggaaccac ggtcaccgtc 360
tcctca 366
<210> 26
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 26
Gln Val Gln Leu Gln Gln Trp Gly Ala Gly Leu Leu Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Tyr Gly Gly Ser Phe Ser Ile His
20 25 30
His Trp Thr Trp Ile Arg His Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Asn His Arg Gly Ser Thr Asn Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Ile Asp Thr Ser Lys Asn Gln Phe Ser Leu
65 70 75 80
Lys Leu Ser Ala Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Leu Arg Phe Leu Asp Trp Leu Ser Ser Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 27
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 27
ggtggatcct tcagtattca tcac 24
<210> 28
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 28
Gly Gly Ser Phe Ser Ile His His
1 5
<210> 29
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 29
atcaatcatc gtggaagcac c 21
<210> 30
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 30
Ile Asn His Arg Gly Ser Thr
1 5
<210> 31
<211> 48
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 31
gcgagaggct tacgattttt ggactggtta tcgtcctact ttgactac 48
<210> 32
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 32
Ala Arg Gly Leu Arg Phe Leu Asp Trp Leu Ser Ser Tyr Phe Asp Tyr
1 5 10 15
<210> 33
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 33
gacatccagt tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcgagtca gggcattagc gattatttag cctggtatca gcagaaacca 120
gggaaagttc ctaacctcct gatctatgct gcgtccgctt tacaatcagg ggtcccatct 180
cgtttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaggatgttg caacttatta ctgtcaaaat tataacactg ccccgctcac tttcggcggg 300
gggaccaagg tggaaatcaa a 321
<210> 34
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 34
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Asp Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Asn Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ala Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Thr Tyr Tyr Cys Gln Asn Tyr Asn Thr Ala Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 35
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 35
cagggcatta gcgattat 18
<210> 36
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 36
Gln Gly Ile Ser Asp Tyr
1 5
<210> 37
<211> 9
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 37
gctgcgtcc 9
<210> 38
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 38
Ala Ala Ser
1
<210> 39
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 39
caaaattata acactgcccc gctcact 27
<210> 40
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 40
Gln Asn Tyr Asn Thr Ala Pro Leu Thr
1 5
<210> 41
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 41
caggtgcagc tgcaggagtc gggcgcagga ctgttgaagc cttcggagac cctgtccctc 60
acctgcactg tctatggtgg atccttcagt attcatcact ggacctggat ccgccatccc 120
ccagggaagg ggctggagtg gattggggag atcaatcatc gtggaagcac caactacaac 180
ccgtccctca agagtcgagt caccatatca atagacacgt ccaagaacca gttctccctg 240
aagctgagcg ctgtgaccgc cgcggacacg gctgtatatt actgtgcgag aggcttacga 300
tttttggact ggttatcgtc ctactttgac tactggggcc agggaaccct ggtcactgtc 360
tcctca 366
<210> 42
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 42
Gln Val Gln Leu Gln Glu Ser Gly Ala Gly Leu Leu Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Tyr Gly Gly Ser Phe Ser Ile His
20 25 30
His Trp Thr Trp Ile Arg His Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Asn His Arg Gly Ser Thr Asn Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Ile Asp Thr Ser Lys Asn Gln Phe Ser Leu
65 70 75 80
Lys Leu Ser Ala Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Leu Arg Phe Leu Asp Trp Leu Ser Ser Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 43
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 43
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcgagtca gggcattagc gattatttag cctggtatca gcagaaacca 120
gggaaagttc ctaacctcct gatctatgct gcgtccgctt tacaatcagg ggtcccatct 180
cgtttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaggatgttg caacttatta ctgtcaaaat tataacactg ccccgctcac tttcggcggg 300
gggaccaagg tggaaatcaa a 321
<210> 44
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 44
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Asp Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Asn Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ala Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Thr Tyr Tyr Cys Gln Asn Tyr Asn Thr Ala Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 45
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 45
caggtgcagc tacagcagtg gggcgcagga ctgttgaagc cttcggagac cctgtccctc 60
acctgcgctg tctatggtgg atccttcagt attcatcact ggagctggat ccgccagccc 120
ccagggaagg ggctggagtg gattggggaa atcaatcatc gtggaagcac caactacaac 180
ccgtccctca agagtcgagt caccatatca gtagacacgt ccaagaacca gttctccctg 240
aagctgagct ctgtgaccgc cgcggacacg gctgtgtatt actgtgcgag aggcttacga 300
tttttggact ggttatcgtc ctactttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 46
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 46
Gln Val Gln Leu Gln Gln Trp Gly Ala Gly Leu Leu Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Val Tyr Gly Gly Ser Phe Ser Ile His
20 25 30
His Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Asn His Arg Gly Ser Thr Asn Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Leu Arg Phe Leu Asp Trp Leu Ser Ser Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 47
<211> 322
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 47
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcgagtca gggcattagc gattatttag cctggtatca gcagaaacca 120
gggaaagttc ctaagctcct gatctatgct gcgtccactt tgcaatcagg ggtcccatct 180
cggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagatgttg caacttatta ctgtcaaaat tataacactg ccccgctcac tttcggcgga 300
gggaccaagg tggagatcaa ac 322
<210> 48
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 48
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Asp Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Thr Tyr Tyr Cys Gln Asn Tyr Asn Thr Ala Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 49
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 49
caggtgcagc tggtgcagtc tgggggaggc ttggtgcagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cactttcagt agctacgaca tgcactgggt ccgccaagtt 120
gcaggaaaag gtctggagtg ggtctcagcc attggtactg ctggtgacac atactatcca 180
gtctccgtga agggccgatt caccatctct agagaaaatg ccaagaactc cttgtatctt 240
cacatgaaca gcctgagagc cggggacacg gctgtgtatt tctgtgcaag aggagacagt 300
agaaactact acgttgggga ctactttgac tactggggcc agggaaccac ggtcaccgtc 360
tcctca 366
<210> 50
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 50
Gln Val Gln Leu Val Gln Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Val Ala Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Pro Val Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
His Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Phe Cys Ala
85 90 95
Arg Gly Asp Ser Arg Asn Tyr Tyr Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 51
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 51
ggattcactt tcagtagcta cgac 24
<210> 52
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 52
Gly Phe Thr Phe Ser Ser Tyr Asp
1 5
<210> 53
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 53
attggtactg ctggtgacac a 21
<210> 54
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 54
Ile Gly Thr Ala Gly Asp Thr
1 5
<210> 55
<211> 48
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 55
gcaagaggag acagtagaaa ctactacgtt ggggactact ttgactac 48
<210> 56
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 56
Ala Arg Gly Asp Ser Arg Asn Tyr Tyr Val Gly Asp Tyr Phe Asp Tyr
1 5 10 15
<210> 57
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 57
gacatcgtga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt aggtggttgg cctggtatca gcaaaaacca 120
ggaaaagccc ctaaggtcct gatctataag gcgtctaatt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag tataatagtt attctcggac gttcggccaa 300
gggaccaagg tggagatcaa a 321
<210> 58
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 58
Asp Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Arg Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asn Ser Tyr Ser Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 59
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 59
cagagtatta gtaggtgg 18
<210> 60
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 60
Gln Ser Ile Ser Arg Trp
1 5
<210> 61
<211> 9
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 61
aaggcgtct 9
<210> 62
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 62
Lys Ala Ser
1
<210> 63
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 63
caacagtata atagttattc tcggacg 27
<210> 64
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 64
Gln Gln Tyr Asn Ser Tyr Ser Arg Thr
1 5
<210> 65
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 65
gaggtgcagc tggtggagtc tgggggaggc ttggtgcagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cactttcagt agctacgaca tgcactgggt ccgccaagtt 120
gcaggaaaag gtctggagtg ggtctcagcc attggtactg ctggtgacac atactatcca 180
gtctccgtga agggccgatt caccatctct agagaaaatg ccaagaactc cttgtatctt 240
cacatgaaca gcctgagagc cggggacacg gctgtgtatt tctgtgcaag aggagacagt 300
agaaactact acgttgggga ctactttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 66
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 66
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Val Ala Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Pro Val Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
His Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Phe Cys Ala
85 90 95
Arg Gly Asp Ser Arg Asn Tyr Tyr Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 67
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 67
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt aggtggttgg cctggtatca gcaaaaacca 120
ggaaaagccc ctaaggtcct gatctataag gcgtctaatt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag tataatagtt attctcggac gttcggccaa 300
gggaccaagg tggaaatcaa a 321
<210> 68
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 68
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Arg Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asn Ser Tyr Ser Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 69
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 69
gaggtgcagc tggtggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cactttcagt agctacgaca tgcactgggt ccgccaagct 120
acaggaaaag gtctggagtg ggtctcagct attggtactg ctggtgacac atactatcca 180
ggctccgtga agggccgatt caccatctcc agagaaaatg ccaagaactc cttgtatctt 240
caaatgaaca gcctgagagc cggggacacg gctgtgtatt actgtgcaag aggagacagt 300
agaaactact acgttgggga ctactttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 70
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 70
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Ala Thr Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Pro Gly Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Arg Asn Tyr Tyr Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 71
<211> 322
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 71
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt aggtggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctataag gcgtctagtt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag tataatagtt attctcggac gttcggccaa 300
gggaccaagg tggaaatcaa ac 322
<210> 72
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 72
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Arg Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asn Ser Tyr Ser Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 73
<211> 378
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 73
gaggtgcagc tggtgcagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agttatggca tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg gatggcagtt atatcatttg atggaggtaa taaaaataat 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agctgaggac acggctgtgt attattgtgc gaaagagggc 300
gatagaagtg gtcaccctta cttctactat tacggtttgg acgtctgggg ccaagggacc 360
acggtcaccg tctcctca 378
<210> 74
<211> 126
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 74
Glu Val Gln Leu Val Gln Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Ala Val Ile Ser Phe Asp Gly Gly Asn Lys Asn Asn Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Glu Gly Asp Arg Ser Gly His Pro Tyr Phe Tyr Tyr Tyr Gly
100 105 110
Leu Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 75
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 75
ggattcacct tcagtagtta tggc 24
<210> 76
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 76
Gly Phe Thr Phe Ser Ser Tyr Gly
1 5
<210> 77
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 77
atatcatttg atggaggtaa taaa 24
<210> 78
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 78
Ile Ser Phe Asp Gly Gly Asn Lys
1 5
<210> 79
<211> 57
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 79
gcgaaagagg gcgatagaag tggtcaccct tacttctact attacggttt ggacgtc 57
<210> 80
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 80
Ala Lys Glu Gly Asp Arg Ser Gly His Pro Tyr Phe Tyr Tyr Tyr Gly
1 5 10 15
Leu Asp Val
<210> 81
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 81
gccatccaga tgacccagtc tccatccttc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgct gggccagtca gggcattagc agttatttag cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccactt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgagacagaa ttcactctca caatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacag cttcatagtt accctctcac tttcggcgga 300
gggaccaagg tggaaatcaa a 321
<210> 82
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 82
Ala Ile Gln Met Thr Gln Ser Pro Ser Phe Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Trp Ala Ser Gln Gly Ile Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Glu Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Leu His Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 83
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 83
cagggcatta gcagttat 18
<210> 84
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 84
Gln Gly Ile Ser Ser Tyr
1 5
<210> 85
<211> 9
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 85
gctgcatcc 9
<210> 86
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 86
Ala Ala Ser
1
<210> 87
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 87
caacagcttc atagttaccc tctcact 27
<210> 88
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 88
Gln Gln Leu His Ser Tyr Pro Leu Thr
1 5
<210> 89
<211> 375
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 89
caggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agttatggca tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg gatggcagtt atatcatttg atggaggtaa taaaaataat 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agctgaggac acggctgtgt attattgtgc gaaagagggc 300
gatagaagtg gtcaccctta cttctactat tacggtttgg acgtctgggg ccaagggacc 360
acggtcaccg tctcc 375
<210> 90
<211> 125
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 90
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Ala Val Ile Ser Phe Asp Gly Gly Asn Lys Asn Asn Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Glu Gly Asp Arg Ser Gly His Pro Tyr Phe Tyr Tyr Tyr Gly
100 105 110
Leu Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
<210> 91
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 91
gacatccagt tgacccagtc tccatccttc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgct gggccagtca gggcattagc agttatttag cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccactt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgagacagaa ttcactctca caatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacag cttcatagtt accctctcac tttcggcgga 300
gggaccaagg tggaaatcaa a 321
<210> 92
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 92
Asp Ile Gln Leu Thr Gln Ser Pro Ser Phe Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Trp Ala Ser Gln Gly Ile Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Glu Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Leu His Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 93
<211> 376
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 93
caggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agttatggca tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtggcagtt atatcatttg atggaggtaa taaatactat 180
gcagactccg tgaagggccg attcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agctgaggac acggctgtgt attactgtgc gaaagagggc 300
gatagaagtg gtcaccctta cttctactat tacggtttgg acgtctgggg ccaagggacc 360
acggtcaccg tctcct 376
<210> 94
<211> 125
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 94
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Phe Asp Gly Gly Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Glu Gly Asp Arg Ser Gly His Pro Tyr Phe Tyr Tyr Tyr Gly
100 105 110
Leu Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
<210> 95
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 95
gacatccagt tgacccagtc tccatccttc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgca gagccagtca gggcattagc agttatttag cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccactt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tggcacagaa ttcactctca caatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacag cttcatagtt accctctcac tttcggcgga 300
gggaccaagg tggagatcaa a 321
<210> 96
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 96
Asp Ile Gln Leu Thr Gln Ser Pro Ser Phe Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Leu His Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 97
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 97
gaggtgcagc tggtgcagtc tgggggaggc ttagtacagc cgggggggtc cctgcgactc 60
tcctgtgcag cctctggatt caccttcagt cggtacgaca tgcactgggt ccgccaagtg 120
acaggaaaag gtctggaatg ggtatcaggc attggtacag caggtgacac atactatcca 180
ggctccgtga agggccgatt caccatctcc agagaaaatg acaagaactc cctgtatctt 240
caaatgaaca gcctgagagt cggggacacg gctgtttatt actgtgcaag aggagatagt 300
aagaactact acgttgggga ctactttgac tactggggcc agggaaccac ggtcaccgtc 360
tcctca 366
<210> 98
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 98
Glu Val Gln Leu Val Gln Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Arg Tyr
20 25 30
Asp Met His Trp Val Arg Gln Val Thr Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Pro Gly Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Asp Lys Asn Ser Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Val Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Lys Asn Tyr Tyr Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 99
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 99
ggattcacct tcagtcggta cgac 24
<210> 100
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 100
Gly Phe Thr Phe Ser Arg Tyr Asp
1 5
<210> 101
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 101
attggtacag caggtgacac a 21
<210> 102
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 102
Ile Gly Thr Ala Gly Asp Thr
1 5
<210> 103
<211> 48
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 103
gcaagaggag atagtaagaa ctactacgtt ggggactact ttgactac 48
<210> 104
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 104
Ala Arg Gly Asp Ser Lys Asn Tyr Tyr Val Gly Asp Tyr Phe Asp Tyr
1 5 10 15
<210> 105
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 105
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
ttcacttgcc gggccagtca cagtattggt aattggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaaggtcct gatctatgag gcgtctagtt tagaagatgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacaa tatgatactt attttcggac gttcggccaa 300
gggaccaagg tggagatcaa a 321
<210> 106
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 106
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Phe Thr Cys Arg Ala Ser His Ser Ile Gly Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Tyr Glu Ala Ser Ser Leu Glu Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Thr Tyr Phe Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 107
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 107
cacagtattg gtaattgg 18
<210> 108
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 108
His Ser Ile Gly Asn Trp
1 5
<210> 109
<211> 9
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 109
gaggcgtct 9
<210> 110
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 110
Glu Ala Ser
1
<210> 111
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 111
caacaatatg atacttattt tcggacg 27
<210> 112
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 112
Gln Gln Tyr Asp Thr Tyr Phe Arg Thr
1 5
<210> 113
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 113
gaggtgcagc tggtggagtc tgggggaggc ttagtacagc cgggggggtc cctgcgactc 60
tcctgtgcag cctctggatt caccttcagt cggtacgaca tgcactgggt ccgccaagtg 120
acaggaaaag gtctggaatg ggtatcaggc attggtacag caggtgacac atactatcca 180
ggctccgtga agggccgatt caccatctcc agagaaaatg acaagaactc cctgtatctt 240
caaatgaaca gcctgagagt cggggacacg gctgtttatt actgtgcaag aggagatagt 300
aagaactact acgttgggga ctactttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 114
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 114
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Arg Tyr
20 25 30
Asp Met His Trp Val Arg Gln Val Thr Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Pro Gly Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Asp Lys Asn Ser Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Val Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Lys Asn Tyr Tyr Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 115
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 115
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
ttcacttgcc gggccagtca cagtattggt aattggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaaggtcct gatctatgag gcgtctagtt tagaagatgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacaa tatgatactt attttcggac gttcggccaa 300
gggaccaagg tggaaatcaa a 321
<210> 116
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 116
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Phe Thr Cys Arg Ala Ser His Ser Ile Gly Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Tyr Glu Ala Ser Ser Leu Glu Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Thr Tyr Phe Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 117
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 117
gaggtgcagc tggtggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt cggtacgaca tgcactgggt ccgccaagct 120
acaggaaaag gtctggagtg ggtctcagct attggtacag caggtgacac atactatcca 180
ggctccgtga agggccgatt caccatctcc agagaaaatg ccaagaactc cttgtatctt 240
caaatgaaca gcctgagagc cggggacacg gctgtgtatt actgtgcaag aggagatagt 300
aagaactact acgttgggga ctactttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 118
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 118
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Arg Tyr
20 25 30
Asp Met His Trp Val Arg Gln Ala Thr Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Pro Gly Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Lys Asn Tyr Tyr Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 119
<211> 322
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 119
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca cagtattggt aattggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgag gcgtctagtt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacaa tatgatactt attttcggac gttcggccaa 300
gggaccaagg tggaaatcaa ac 322
<210> 120
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 120
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser His Ser Ile Gly Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Glu Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Thr Tyr Phe Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 121
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 121
caggtgcagc tggtggagtc cgggggaggc ttggtccagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt acctacgaca tgcactgggt ccgccaaggt 120
ctaggaaaag gtctggagtg ggtctcagct attggttctg ctggtgacac atactatcca 180
ggctccgtga agggccgctt caccatctcc agagacaatg ccaagagctc cttgtttctt 240
cacatgaaca gcctgagagc cggggacacg gctctatatt actgtgcaag aggagatagt 300
cggaactact tcgttgggga ctactttgac tactggggcc agggaaccac ggtcaccgtc 360
tcctca 366
<210> 122
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 122
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Tyr
20 25 30
Asp Met His Trp Val Arg Gln Gly Leu Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Ser Ala Gly Asp Thr Tyr Tyr Pro Gly Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Ser Ser Leu Phe Leu
65 70 75 80
His Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Leu Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Arg Asn Tyr Phe Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 123
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 123
ggattcacct tcagtaccta cgac 24
<210> 124
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 124
Gly Phe Thr Phe Ser Thr Tyr Asp
1 5
<210> 125
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 125
attggttctg ctggtgacac a 21
<210> 126
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 126
Ile Gly Ser Ala Gly Asp Thr
1 5
<210> 127
<211> 48
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 127
gcaagaggag atagtcggaa ctacttcgtt ggggactact ttgactac 48
<210> 128
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 128
Ala Arg Gly Asp Ser Arg Asn Tyr Phe Val Gly Asp Tyr Phe Asp Tyr
1 5 10 15
<210> 129
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 129
gccatccaga tgacccagtc tccgtccacc ctgtctgcat ctataggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt agttggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaaggtcct gatctataag gcgtctagtt tagaagctgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctacagcct 240
gatgattttg caagttatta ctgccaacag tatagtagtt attctcggac gttcggccaa 300
gggaccaagg tggagatcaa a 321
<210> 130
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 130
Ala Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Ile Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Tyr Lys Ala Ser Ser Leu Glu Ala Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Ser Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Ser Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 131
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 131
cagagtatta gtagttgg 18
<210> 132
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 132
Gln Ser Ile Ser Ser Trp
1 5
<210> 133
<211> 9
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 133
aaggcgtct 9
<210> 134
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 134
Lys Ala Ser
1
<210> 135
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 135
caacagtata gtagttattc tcggacg 27
<210> 136
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 136
Gln Gln Tyr Ser Ser Tyr Ser Arg Thr
1 5
<210> 137
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 137
gaggtgcagc tggtggagtc tgggggaggc ttggtccagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt acctacgaca tgcactgggt ccgccaaggt 120
ctaggaaaag gtctggagtg ggtctcagct attggttctg ctggtgacac atactatcca 180
ggctccgtga agggccgctt caccatctcc agagacaatg ccaagagctc cttgtttctt 240
cacatgaaca gcctgagagc cggggacacg gctctatatt actgtgcaag aggagatagt 300
cggaactact tcgttgggga ctactttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 138
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 138
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Tyr
20 25 30
Asp Met His Trp Val Arg Gln Gly Leu Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Ser Ala Gly Asp Thr Tyr Tyr Pro Gly Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Ser Ser Leu Phe Leu
65 70 75 80
His Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Leu Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Arg Asn Tyr Phe Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 139
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 139
gacatccaga tgacccagtc tccgtccacc ctgtctgcat ctataggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt agttggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaaggtcct gatctataag gcgtctagtt tagaagctgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctacagcct 240
gatgattttg caagttatta ctgccaacag tatagtagtt attctcggac gttcggccaa 300
gggaccaagg tggaaatcaa a 321
<210> 140
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 140
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Ile Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Tyr Lys Ala Ser Ser Leu Glu Ala Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Ser Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Ser Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 141
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 141
gaggtgcagc tggtggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt acctacgaca tgcactgggt ccgccaagct 120
acaggaaaag gtctggagtg ggtctcagct attggttctg ctggtgacac atactatcca 180
ggctccgtga agggccgatt caccatctcc agagaaaatg ccaagaactc cttgtatctt 240
caaatgaaca gcctgagagc cggggacacg gctgtgtatt actgtgcaag aggagatagt 300
cggaactact tcgttgggga ctactttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 142
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 142
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Tyr
20 25 30
Asp Met His Trp Val Arg Gln Ala Thr Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Ser Ala Gly Asp Thr Tyr Tyr Pro Gly Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Arg Asn Tyr Phe Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 143
<211> 322
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 143
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt agttggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctataag gcgtctagtt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag tatagtagtt attctcggac gttcggccaa 300
gggaccaagg tggaaatcaa ac 322
<210> 144
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 144
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Ser Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 145
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 145
caggtgcagc tggtggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgtag cctctggatt caccttcagt agttacgaca tgcactgggt ccgccaactt 120
ccaggaaaag gtctggagtg ggtctcagcc attggtgttg ctggtgacac atactatcca 180
gcctccgtga agggccgatt caccatctct agagaaaatg ccaagaactc cttgtatctt 240
caaatgaaca gcctgagagc cggggacacg gctgtgtatt actgtgcaag aggagacagt 300
gggaactact acgatgggga ctactttgac ttctggggcc agggaaccac ggtcaccgtc 360
tcctca 366
<210> 146
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 146
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Val Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Leu Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Val Ala Gly Asp Thr Tyr Tyr Pro Ala Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Gly Asn Tyr Tyr Asp Gly Asp Tyr Phe Asp Phe Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 147
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 147
ggattcacct tcagtagtta cgac 24
<210> 148
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 148
Gly Phe Thr Phe Ser Ser Tyr Asp
1 5
<210> 149
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 149
attggtgttg ctggtgacac a 21
<210> 150
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 150
Ile Gly Val Ala Gly Asp Thr
1 5
<210> 151
<211> 48
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 151
gcaagaggag acagtgggaa ctactacgat ggggactact ttgacttc 48
<210> 152
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 152
Ala Arg Gly Asp Ser Gly Asn Tyr Tyr Asp Gly Asp Tyr Phe Asp Phe
1 5 10 15
<210> 153
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 153
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattaat aggtggttgg cctggtatca gcaaaaacca 120
gggaaagccc ctaaggtcct gatctataag gcgtctaatt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag tatgatagtt attttcggac gttcggccaa 300
gggaccaagg tggagatcaa a 321
<210> 154
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 154
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Asn Arg Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Ser Tyr Phe Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 155
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 155
cagagtatta ataggtgg 18
<210> 156
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 156
Gln Ser Ile Asn Arg Trp
1 5
<210> 157
<211> 9
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 157
aaggcgtct 9
<210> 158
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 158
Lys Ala Ser
1
<210> 159
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 159
caacagtatg atagttattt tcggacg 27
<210> 160
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 160
Gln Gln Tyr Asp Ser Tyr Phe Arg Thr
1 5
<210> 161
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 161
gaggtgcagc tggtggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgtag cctctggatt caccttcagt agttacgaca tgcactgggt ccgccaactt 120
ccaggaaaag gtctggagtg ggtctcagcc attggtgttg ctggtgacac atactatcca 180
gcctccgtga agggccgatt caccatctct agagaaaatg ccaagaactc cttgtatctt 240
caaatgaaca gcctgagagc cggggacacg gctgtgtatt actgtgcaag aggagacagt 300
gggaactact acgatgggga ctactttgac ttctggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 162
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 162
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Val Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Leu Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Val Ala Gly Asp Thr Tyr Tyr Pro Ala Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Gly Asn Tyr Tyr Asp Gly Asp Tyr Phe Asp Phe Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 163
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 163
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattaat aggtggttgg cctggtatca gcaaaaacca 120
gggaaagccc ctaaggtcct gatctataag gcgtctaatt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag tatgatagtt attttcggac gttcggccaa 300
gggaccaagg tggagatcaa a 321
<210> 164
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 164
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Asn Arg Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Ser Tyr Phe Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 165
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 165
gaggtgcagc tggtggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agttacgaca tgcactgggt ccgccaagct 120
acaggaaaag gtctggagtg ggtctcagct attggtgttg ctggtgacac atactatcca 180
ggctccgtga agggccgatt caccatctcc agagaaaatg ccaagaactc cttgtatctt 240
caaatgaaca gcctgagagc cggggacacg gctgtgtatt actgtgcaag aggagacagt 300
gggaactact acgatgggga ctactttgac ttctggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 166
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 166
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Ala Thr Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Val Ala Gly Asp Thr Tyr Tyr Pro Gly Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Gly Asn Tyr Tyr Asp Gly Asp Tyr Phe Asp Phe Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 167
<211> 322
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 167
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattaat aggtggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctataag gcgtctagtt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag tatgatagtt attttcggac gttcggccaa 300
gggaccaagg tggaaatcaa ac 322
<210> 168
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 168
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Asn Arg Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Ser Tyr Phe Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 169
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 169
gaggtgcagc tggtggagtc tgggggaggc ttggtccagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt acctacgaca tgcactgggt ccgccaagct 120
ccaggaaaag gtctggagtg ggtctcagct attggttctg ctggtgacac atactatcca 180
ggctccgtga agggccgatt caccatctcc agagacaatg ccaagagctc cttgtttctt 240
caaatgaaca gcctgagagc cggggacacg gctctatatt actgtgcaag aggagatagt 300
cggaactact tcgttgggga ctactttgac tactggggcc agggaaccac ggtcaccgtc 360
tcctca 366
<210> 170
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 170
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Tyr
20 25 30
Asp Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Ser Ala Gly Asp Thr Tyr Tyr Pro Gly Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Ser Ser Leu Phe Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Leu Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Arg Asn Tyr Phe Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 171
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 171
ggattcacct tcagtaccta cgac 24
<210> 172
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 172
Gly Phe Thr Phe Ser Thr Tyr Asp
1 5
<210> 173
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 173
attggttctg ctggtgacac a 21
<210> 174
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 174
Ile Gly Ser Ala Gly Asp Thr
1 5
<210> 175
<211> 48
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 175
gcaagaggag atagtcggaa ctacttcgtt ggggactact ttgactac 48
<210> 176
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 176
Ala Arg Gly Asp Ser Arg Asn Tyr Phe Val Gly Asp Tyr Phe Asp Tyr
1 5 10 15
<210> 177
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 177
gacatccagt tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattggt acctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagatcct gatctataag gcgtctagtt tagaaggtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caaattacta ctgccaacag tataatagtt tttatcggac gttcggccaa 300
gggaccaagg tggagatcaa a 321
<210> 178
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 178
Asp Ile Gln Leu Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Gly Thr Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Ile Leu Ile
35 40 45
Tyr Lys Ala Ser Ser Leu Glu Gly Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Asn Tyr Tyr Cys Gln Gln Tyr Asn Ser Phe Tyr Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 179
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 179
cagagtattg gtacctgg 18
<210> 180
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 180
Gln Ser Ile Gly Thr Trp
1 5
<210> 181
<211> 9
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 181
aaggcgtct 9
<210> 182
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 182
Lys Ala Ser
1
<210> 183
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 183
caacagtata atagttttta tcggacg 27
<210> 184
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 184
Gln Gln Tyr Asn Ser Phe Tyr Arg Thr
1 5
<210> 185
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 185
gaggtgcagc tggtggagtc tgggggaggc ttggtccagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt acctacgaca tgcactgggt ccgccaagct 120
ccaggaaaag gtctggagtg ggtctcagct attggttctg ctggtgacac atactatcca 180
ggctccgtga agggccgatt caccatctcc agagacaatg ccaagagctc cttgtttctt 240
caaatgaaca gcctgagagc cggggacacg gctctatatt actgtgcaag aggagatagt 300
cggaactact tcgttgggga ctactttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 186
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 186
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Tyr
20 25 30
Asp Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Ser Ala Gly Asp Thr Tyr Tyr Pro Gly Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Ser Ser Leu Phe Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Leu Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Arg Asn Tyr Phe Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 187
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 187
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattggt acctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagatcct gatctataag gcgtctagtt tagaaggtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caaattacta ctgccaacag tataatagtt tttatcggac gttcggccaa 300
gggaccaagg tggaaatcaa a 321
<210> 188
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 188
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Gly Thr Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Ile Leu Ile
35 40 45
Tyr Lys Ala Ser Ser Leu Glu Gly Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Asn Tyr Tyr Cys Gln Gln Tyr Asn Ser Phe Tyr Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 189
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 189
gaggtgcagc tggtggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt acctacgaca tgcactgggt ccgccaagct 120
acaggaaaag gtctggagtg ggtctcagct attggttctg ctggtgacac atactatcca 180
ggctccgtga agggccgatt caccatctcc agagaaaatg ccaagaactc cttgtatctt 240
caaatgaaca gcctgagagc cggggacacg gctgtgtatt actgtgcaag aggagatagt 300
cggaactact tcgttgggga ctactttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 190
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 190
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Tyr
20 25 30
Asp Met His Trp Val Arg Gln Ala Thr Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Ser Ala Gly Asp Thr Tyr Tyr Pro Gly Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Arg Asn Tyr Phe Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 191
<211> 322
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 191
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattggt acctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctataag gcgtctagtt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag tataatagtt tttatcggac gttcggccaa 300
gggaccaagg tggaaatcaa ac 322
<210> 192
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 192
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Gly Thr Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asn Ser Phe Tyr Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 193
<211> 378
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 193
caggtgcagc tggtgcagtc tgggggaggc ttggtacagc cgggggggtc cctgagactc 60
tcctgtgcag cctctgaatt catttttagc agctatgcca tgaactgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagtc attagtggta gtggtgatag caaatactac 180
gcagactccg tgaagggccg attcaccatc tccagagaca attccaagaa cacactgtat 240
ctgcaaatga acagcctgag agtcgaggac acggccgtgt attactgtgc gaaagatggg 300
aaggacaggt atggttttta ctacaacttc tacggtatgg acgtctgggg ccaagggacc 360
acggtcaccg tctcctca 378
<210> 194
<211> 126
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 194
Gln Val Gln Leu Val Gln Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Glu Phe Ile Phe Ser Ser Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Ser Gly Ser Gly Asp Ser Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Val Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Asp Gly Lys Asp Arg Tyr Gly Phe Tyr Tyr Asn Phe Tyr Gly
100 105 110
Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 195
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 195
gaattcattt ttagcagcta tgcc 24
<210> 196
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 196
Glu Phe Ile Phe Ser Ser Tyr Ala
1 5
<210> 197
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 197
attagtggta gtggtgatag caaa 24
<210> 198
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 198
Ile Ser Gly Ser Gly Asp Ser Lys
1 5
<210> 199
<211> 57
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 199
gcgaaagatg ggaaggacag gtatggtttt tactacaact tctacggtat ggacgtc 57
<210> 200
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 200
Ala Lys Asp Gly Lys Asp Arg Tyr Gly Phe Tyr Tyr Asn Phe Tyr Gly
1 5 10 15
Met Asp Val
<210> 201
<211> 324
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 201
gcacatccga tgacccagtc tccatccttc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgct gggccagtca gggcataagc agttatttag cctggtatca gcaaaaacca 120
gggaaagccc ctaagatcct gatctatgct gcatccactt tgcaaagtgg ggtcccatca 180
aggttcagcg ccagtgggtc tgggacagaa ttcactctca caatcagcag cctgcagcct 240
gaagattttg caacttatca ctgtcaacag cttaatagtt acccattcac tttcggccct 300
gggaccaagg tggaaatcaa acga 324
<210> 202
<211> 108
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 202
Ala His Pro Met Thr Gln Ser Pro Ser Phe Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Trp Ala Ser Gln Gly Ile Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Ile Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Ala
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr His Cys Gln Gln Leu Asn Ser Tyr Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Glu Ile Lys Arg
100 105
<210> 203
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 203
cagggcataa gcagttat 18
<210> 204
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 204
Gln Gly Ile Ser Ser Tyr
1 5
<210> 205
<211> 9
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 205
gctgcatcc 9
<210> 206
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 206
Ala Ala Ser
1
<210> 207
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 207
caacagctta atagttaccc attcact 27
<210> 208
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 208
Gln Gln Leu Asn Ser Tyr Pro Phe Thr
1 5
<210> 209
<211> 375
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 209
gaggtgcagc tggtggagtc tgggggaggc ttggtacagc cgggggggtc cctgagactc 60
tcctgtgcag cctctgaatt catttttagc agctatgcca tgaactgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagtc attagtggta gtggtgatag caaatactac 180
gcagactccg tgaagggccg attcaccatc tccagagaca attccaagaa cacactgtat 240
ctgcaaatga acagcctgag agtcgaggac acggccgtgt attactgtgc gaaagatggg 300
aaggacaggt atggttttta ctacaacttc tacggtatgg acgtctgggg ccaagggacc 360
acggtcaccg tctcc 375
<210> 210
<211> 125
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 210
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Glu Phe Ile Phe Ser Ser Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Ser Gly Ser Gly Asp Ser Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Val Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Asp Gly Lys Asp Arg Tyr Gly Phe Tyr Tyr Asn Phe Tyr Gly
100 105 110
Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
<210> 211
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 211
gacatccaga tgacccagtc tccatccttc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgct gggccagtca gggcataagc agttatttag cctggtatca gcaaaaacca 120
gggaaagccc ctaagatcct gatctatgct gcatccactt tgcaaagtgg ggtcccatca 180
aggttcagcg ccagtgggtc tgggacagaa ttcactctca caatcagcag cctgcagcct 240
gaagattttg caacttatca ctgtcaacag cttaatagtt acccattcac tttcggccct 300
gggaccaaag tggatatcaa a 321
<210> 212
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 212
Asp Ile Gln Met Thr Gln Ser Pro Ser Phe Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Trp Ala Ser Gln Gly Ile Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Ile Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Ala
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr His Cys Gln Gln Leu Asn Ser Tyr Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 213
<211> 376
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 213
gaggtgcagc tggtggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctgaatt catttttagc agctatgcca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggta gtggtgatag caaatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agccgaggac acggccgtat attactgtgc gaaagatggg 300
aaggacaggt atggttttta ctacaacttc tacggtatgg acgtctgggg ccaagggacc 360
acggtcaccg tctcct 376
<210> 214
<211> 125
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 214
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Glu Phe Ile Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Asp Ser Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Asp Gly Lys Asp Arg Tyr Gly Phe Tyr Tyr Asn Phe Tyr Gly
100 105 110
Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
<210> 215
<211> 322
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 215
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gggcataagc agttatttag gctggtatca gcagaaacca 120
gggaaagccc ctaagcgcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca caatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacag cttaatagtt acccattcac tttcggccct 300
gggaccaaag tggatatcaa ac 322
<210> 216
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 216
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Ser Tyr
20 25 30
Leu Gly Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Leu Asn Ser Tyr Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 217
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 217
gaggtgcagc tggtgcagtc tgggggagac ttggtacagt ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt tcctacgaca tgcactgggt ccgccaagtt 120
aaaggaaaag gtctggagtg ggtctcagct attggaactg ctggtgacac atactatcaa 180
ggctccgtga agggccgatt caccatctcc agagaaaatg ccaagaactc cttgtttctt 240
caaatgaaca gcctgagagc cggggacacg gctgtatatt actgtgcaag aggagatagt 300
agaaactact tcgttgggga ctactttgac tactggggcc agggaaccac ggtcaccgtc 360
tcctca 366
<210> 218
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 218
Glu Val Gln Leu Val Gln Ser Gly Gly Asp Leu Val Gln Ser Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Val Lys Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Gln Gly Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Phe Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Arg Asn Tyr Phe Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 219
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 219
ggattcacct tcagttccta cgac 24
<210> 220
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 220
Gly Phe Thr Phe Ser Ser Tyr Asp
1 5
<210> 221
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 221
attggaactg ctggtgacac a 21
<210> 222
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 222
Ile Gly Thr Ala Gly Asp Thr
1 5
<210> 223
<211> 48
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 223
gcaagaggag atagtagaaa ctacttcgtt ggggactact ttgactac 48
<210> 224
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 224
Ala Arg Gly Asp Ser Arg Asn Tyr Phe Val Gly Asp Tyr Phe Asp Tyr
1 5 10 15
<210> 225
<211> 324
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 225
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt aactggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaaggtcct gatctataag gcgtctaatt tagaaggtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccataagcaa cctgcagcct 240
gatgattttg caacttatta ctgccaacag tataatagtt attctcggac gttcggccaa 300
gggaccaagg tggaaatcaa acga 324
<210> 226
<211> 108
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 226
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu Glu Gly Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Asn Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asn Ser Tyr Ser Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg
100 105
<210> 227
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 227
cagagtatta gtaactgg 18
<210> 228
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 228
Gln Ser Ile Ser Asn Trp
1 5
<210> 229
<211> 9
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 229
aaggcgtct 9
<210> 230
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 230
Lys Ala Ser
1
<210> 231
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 231
caacagtata atagttattc tcggacg 27
<210> 232
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 232
Gln Gln Tyr Asn Ser Tyr Ser Arg Thr
1 5
<210> 233
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 233
gaggtgcagc tggtggagtc tgggggagac ttggtacagt ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt tcctacgaca tgcactgggt ccgccaagtt 120
aaaggaaaag gtctggagtg ggtctcagct attggaactg ctggtgacac atactatcaa 180
ggctccgtga agggccgatt caccatctcc agagaaaatg ccaagaactc cttgtttctt 240
caaatgaaca gcctgagagc cggggacacg gctgtatatt actgtgcaag aggagatagt 300
agaaactact tcgttgggga ctactttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 234
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 234
Glu Val Gln Leu Val Glu Ser Gly Gly Asp Leu Val Gln Ser Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Val Lys Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Gln Gly Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Phe Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Arg Asn Tyr Phe Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 235
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 235
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt aactggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaaggtcct gatctataag gcgtctaatt tagaaggtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccataagcaa cctgcagcct 240
gatgattttg caacttatta ctgccaacag tataatagtt attctcggac gttcggccaa 300
gggaccaagg tggaaatcaa a 321
<210> 236
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 236
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu Glu Gly Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Asn Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asn Ser Tyr Ser Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 237
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 237
gaggtgcagc tggtggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt tcctacgaca tgcactgggt ccgccaagct 120
acaggaaaag gtctggagtg ggtctcagct attggaactg ctggtgacac atactatcca 180
ggctccgtga agggccgatt caccatctcc agagaaaatg ccaagaactc cttgtatctt 240
caaatgaaca gcctgagagc cggggacacg gctgtgtatt actgtgcaag aggagatagt 300
agaaactact tcgttgggga ctactttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 238
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 238
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Ala Thr Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Pro Gly Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Arg Asn Tyr Phe Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 239
<211> 322
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 239
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt aactggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctataag gcgtctagtt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag tataatagtt attctcggac gttcggccaa 300
gggaccaagg tggaaatcaa ac 322
<210> 240
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 240
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asn Ser Tyr Ser Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 241
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 241
caggtgcagc tggtgcagtc tgggggaggc ttggtgcagc ctggggggtc cctgagactc 60
tcctgtgtag cctctggatt caccttcagt agctacgaca tgcactgggt ccgccaagtt 120
gcaggaaaag gtctggagtg ggtcgcagcc attggtactg ctggtgacac atactatcca 180
gtctccgtga agggccgatt caccatctct agagaaaatg ccaagaactc cttgtctctt 240
cacatgaaca gcctgagagc cggggacacg gctgtgtatt actgtgcaag aggagacagt 300
agaaactact acgttgggga ctactttgac tactggggcc agggaaccac ggtcaccgtc 360
tcctca 366
<210> 242
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 242
Gln Val Gln Leu Val Gln Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Val Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Val Ala Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Ala Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Pro Val Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Ser Leu
65 70 75 80
His Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Arg Asn Tyr Tyr Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 243
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 243
ggattcacct tcagtagcta cgac 24
<210> 244
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 244
Gly Phe Thr Phe Ser Ser Tyr Asp
1 5
<210> 245
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 245
attggtactg ctggtgacac a 21
<210> 246
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 246
Ile Gly Thr Ala Gly Asp Thr
1 5
<210> 247
<211> 48
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 247
gcaagaggag acagtagaaa ctactacgtt ggggactact ttgactac 48
<210> 248
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 248
Ala Arg Gly Asp Ser Arg Asn Tyr Tyr Val Gly Asp Tyr Phe Asp Tyr
1 5 10 15
<210> 249
<211> 324
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 249
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt aggtggttgg cctggtatca gcaaaaacca 120
gggaaagccc ctaaggtcct gatctataag gcgtctagtt taaaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagtct 240
gatgattttg caacttatta ctgccaacag tataatagtt attctcggac gttcggccaa 300
gggaccaagg tggagatcaa acga 324
<210> 250
<211> 108
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 250
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Arg Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Tyr Lys Ala Ser Ser Leu Lys Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Ser
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asn Ser Tyr Ser Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg
100 105
<210> 251
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 251
cagagtatta gtaggtgg 18
<210> 252
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 252
Gln Ser Ile Ser Arg Trp
1 5
<210> 253
<211> 9
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 253
aaggcgtct 9
<210> 254
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 254
Lys Ala Ser
1
<210> 255
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 255
caacagtata atagttattc tcggacg 27
<210> 256
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 256
Gln Gln Tyr Asn Ser Tyr Ser Arg Thr
1 5
<210> 257
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 257
gaggtgcagc tggtggagtc tgggggaggc ttggtgcagc ctggggggtc cctgagactc 60
tcctgtgtag cctctggatt caccttcagt agctacgaca tgcactgggt ccgccaagtt 120
gcaggaaaag gtctggagtg ggtcgcagcc attggtactg ctggtgacac atactatcca 180
gtctccgtga agggccgatt caccatctct agagaaaatg ccaagaactc cttgtctctt 240
cacatgaaca gcctgagagc cggggacacg gctgtgtatt actgtgcaag aggagacagt 300
agaaactact acgttgggga ctactttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 258
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 258
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Val Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Val Ala Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Ala Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Pro Val Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Ser Leu
65 70 75 80
His Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Arg Asn Tyr Tyr Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 259
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 259
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt aggtggttgg cctggtatca gcaaaaacca 120
gggaaagccc ctaaggtcct gatctataag gcgtctagtt taaaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagtct 240
gatgattttg caacttatta ctgccaacag tataatagtt attctcggac gttcggccaa 300
gggaccaagg tggaaatcaa a 321
<210> 260
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 260
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Arg Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Tyr Lys Ala Ser Ser Leu Lys Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Ser
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asn Ser Tyr Ser Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 261
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 261
gaggtgcagc tggtggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agctacgaca tgcactgggt ccgccaagct 120
acaggaaaag gtctggagtg ggtctcagct attggtactg ctggtgacac atactatcca 180
ggctccgtga agggccgatt caccatctcc agagaaaatg ccaagaactc cttgtatctt 240
caaatgaaca gcctgagagc cggggacacg gctgtgtatt actgtgcaag aggagacagt 300
agaaactact acgttgggga ctactttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 262
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 262
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Ala Thr Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Pro Gly Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Arg Asn Tyr Tyr Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 263
<211> 322
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 263
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt aggtggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctataag gcgtctagtt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag tataatagtt attctcggac gttcggccaa 300
gggaccaagg tggaaatcaa ac 322
<210> 264
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 264
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Arg Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asn Ser Tyr Ser Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 265
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 265
caggtgcagc tggtacagtc tgggggaggc ttggtgcagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agctacgaca tgcactgggt ccgccaagtt 120
gcaggaaaag gtctggagtg ggtctcagcc attggtactg ctggtgacac atactatcca 180
gtctccgtga agggccgatt caccatctct agagaaaatg ccaagaactc cttgtatctt 240
cacatgaaca gcctgagagc cggggatacg gctgtgtatt actgtgcaag aggagacagt 300
agaaactact acgttgggga ctactttgac tactggggcc agggaaccac ggtcaccgtc 360
tcctca 366
<210> 266
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 266
Gln Val Gln Leu Val Gln Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Val Ala Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Pro Val Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
His Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Arg Asn Tyr Tyr Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 267
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 267
ggattcacct tcagtagcta cgac 24
<210> 268
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 268
Gly Phe Thr Phe Ser Ser Tyr Asp
1 5
<210> 269
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 269
attggtactg ctggtgacac a 21
<210> 270
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 270
Ile Gly Thr Ala Gly Asp Thr
1 5
<210> 271
<211> 48
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 271
gcaagaggag acagtagaaa ctactacgtt ggggactact ttgactac 48
<210> 272
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 272
Ala Arg Gly Asp Ser Arg Asn Tyr Tyr Val Gly Asp Tyr Phe Asp Tyr
1 5 10 15
<210> 273
<211> 324
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 273
gacatccagt tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt aggtggttgg cctggtatca gcaaaaacca 120
gggaaagccc ctaaggtcct gatctataag gcgtctaatt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag tataatactt attctcggac gttcggccaa 300
gggaccaagg tggaaatcaa acga 324
<210> 274
<211> 108
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 274
Asp Ile Gln Leu Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Arg Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asn Thr Tyr Ser Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg
100 105
<210> 275
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 275
cagagtatta gtaggtgg 18
<210> 276
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 276
Gln Ser Ile Ser Arg Trp
1 5
<210> 277
<211> 9
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 277
aaggcgtct 9
<210> 278
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 278
Lys Ala Ser
1
<210> 279
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 279
caacagtata atacttattc tcggacg 27
<210> 280
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 280
Gln Gln Tyr Asn Thr Tyr Ser Arg Thr
1 5
<210> 281
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 281
gaggtgcagc tggtggagtc tgggggaggc ttggtgcagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agctacgaca tgcactgggt ccgccaagtt 120
gcaggaaaag gtctggagtg ggtctcagcc attggtactg ctggtgacac atactatcca 180
gtctccgtga agggccgatt caccatctct agagaaaatg ccaagaactc cttgtatctt 240
cacatgaaca gcctgagagc cggggatacg gctgtgtatt actgtgcaag aggagacagt 300
agaaactact acgttgggga ctactttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 282
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 282
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Val Ala Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Pro Val Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
His Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Arg Asn Tyr Tyr Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 283
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 283
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt aggtggttgg cctggtatca gcaaaaacca 120
gggaaagccc ctaaggtcct gatctataag gcgtctaatt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag tataatactt attctcggac gttcggccaa 300
gggaccaagg tggaaatcaa a 321
<210> 284
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 284
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Arg Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asn Thr Tyr Ser Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 285
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 285
gaggtgcagc tggtggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agctacgaca tgcactgggt ccgccaagct 120
acaggaaaag gtctggagtg ggtctcagct attggtactg ctggtgacac atactatcca 180
ggctccgtga agggccgatt caccatctcc agagaaaatg ccaagaactc cttgtatctt 240
caaatgaaca gcctgagagc cggggacacg gctgtgtatt actgtgcaag aggagacagt 300
agaaactact acgttgggga ctactttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 286
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 286
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Ala Thr Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Pro Gly Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Arg Asn Tyr Tyr Val Gly Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 287
<211> 322
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 287
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt aggtggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctataag gcgtctagtt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag tataatactt attctcggac gttcggccaa 300
gggaccaagg tggaaatcaa ac 322
<210> 288
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 288
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Arg Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asn Thr Tyr Ser Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 289
<211> 363
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 289
caggtgcagc tgcaggagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgtag cgtctggatt caccttcagc agctatggca tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg gatggcagtt atttggtatg atggaagtaa taagtacttt 180
gcagactccg tgaaggaccg attcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acactctgag acctgacgac acggctgtgt attactgtgt gaaggcggac 300
gcccccctcc tgatctatgg tgtggacgtc tggggccaag ggaccacggt caccgtctcc 360
tca 363
<210> 290
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 290
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Val Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Asn Lys Tyr Phe Ala Asp Ser Val
50 55 60
Lys Asp Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Thr Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Ala Asp Ala Pro Leu Leu Ile Tyr Gly Val Asp Val Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 291
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 291
ggattcacct tcagcagcta tggc 24
<210> 292
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 292
Gly Phe Thr Phe Ser Ser Tyr Gly
1 5
<210> 293
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 293
atttggtatg atggaagtaa taag 24
<210> 294
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 294
Ile Trp Tyr Asp Gly Ser Asn Lys
1 5
<210> 295
<211> 42
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 295
gtgaaggcgg acgcccccct cctgatctat ggtgtggacg tc 42
<210> 296
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 296
Val Lys Ala Asp Ala Pro Leu Leu Ile Tyr Gly Val Asp Val
1 5 10
<210> 297
<211> 324
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 297
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gggcattaga aatgatttag gctggtatca agagaaacca 120
ggaaaagccc ctaagcgcct gatctatttt gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca caatcagcag cctgcagcct 240
gaagattttg caacgtatta ctgtctacag cataatagtt acccttacac ttttggccag 300
gggaccaagc tggagatcaa acga 324
<210> 298
<211> 108
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 298
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Arg Asn Asp
20 25 30
Leu Gly Trp Tyr Gln Glu Lys Pro Gly Lys Ala Pro Lys Arg Leu Ile
35 40 45
Tyr Phe Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln His Asn Ser Tyr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg
100 105
<210> 299
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 299
cagggcatta gaaatgat 18
<210> 300
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 300
Gln Gly Ile Arg Asn Asp
1 5
<210> 301
<211> 9
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 301
tttgcatcc 9
<210> 302
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 302
Phe Ala Ser
1
<210> 303
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 303
ctacagcata atagttaccc ttacact 27
<210> 304
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 304
Leu Gln His Asn Ser Tyr Pro Tyr Thr
1 5
<210> 305
<211> 360
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 305
caggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgtag cgtctggatt caccttcagc agctatggca tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg gatggcagtt atttggtatg atggaagtaa taagtacttt 180
gcagactccg tgaaggaccg attcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acactctgag acctgacgac acggctgtgt attactgtgt gaaggcggac 300
gcccccctcc tgatctatgg tgtggacgtc tggggccaag ggaccacggt caccgtctcc 360
<210> 306
<211> 120
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 306
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Val Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Asn Lys Tyr Phe Ala Asp Ser Val
50 55 60
Lys Asp Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Thr Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Ala Asp Ala Pro Leu Leu Ile Tyr Gly Val Asp Val Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser
115 120
<210> 307
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 307
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gggcattaga aatgatttag gctggtatca agagaaacca 120
ggaaaagccc ctaagcgcct gatctatttt gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca caatcagcag cctgcagcct 240
gaagattttg caacgtatta ctgtctacag cataatagtt acccttacac ttttggccag 300
gggaccaagc tggagatcaa a 321
<210> 308
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 308
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Arg Asn Asp
20 25 30
Leu Gly Trp Tyr Gln Glu Lys Pro Gly Lys Ala Pro Lys Arg Leu Ile
35 40 45
Tyr Phe Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln His Asn Ser Tyr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 309
<211> 361
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 309
caggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cgtctggatt caccttcagc agctatggca tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtggcagtt atttggtatg atggaagtaa taagtactat 180
gcagactccg tgaagggccg attcaccatc tccagagaca actccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agccgaggac acggctgtgt attactgtgt gaaggcggac 300
gcccccctcc tgatctatgg tgtggacgtc tggggccaag ggaccacggt caccgtctcc 360
t 361
<210> 310
<211> 120
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 310
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Ala Asp Ala Pro Leu Leu Ile Tyr Gly Val Asp Val Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser
115 120
<210> 311
<211> 322
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 311
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gggcattaga aatgatttag gctggtatca gcagaaacca 120
gggaaagccc ctaagcgcct gatctatttt gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca caatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtctacag cataatagtt acccttacac ttttggccag 300
gggaccaagc tggagatcaa ac 322
<210> 312
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 312
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Arg Asn Asp
20 25 30
Leu Gly Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Arg Leu Ile
35 40 45
Tyr Phe Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln His Asn Ser Tyr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 313
<211> 357
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 313
caggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cgtctggatt caccttcagg agttatggca tacactgggt ccgccaggct 120
ccaggcaagg gactggagtg ggtggcactt atattatatg atggaagtag tgaggactat 180
gcagactccg tgaagggccg attcaccatc tccagagaca attccaagaa tatggtgttt 240
ctgcaaatga acagcctgag agtcgaggac acggctgtct attactgtgc gagagattta 300
ttggcaattg gctggttcga ccgctggggc cagggaaccc tggtcaccgt ctcctca 357
<210> 314
<211> 119
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 314
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Arg Ser Tyr
20 25 30
Gly Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Leu Ile Leu Tyr Asp Gly Ser Ser Glu Asp Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Met Val Phe
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Val Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Leu Leu Ala Ile Gly Trp Phe Asp Arg Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 315
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 315
ggattcacct tcaggagtta tggc 24
<210> 316
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 316
Gly Phe Thr Phe Arg Ser Tyr Gly
1 5
<210> 317
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 317
atattatatg atggaagtag tgag 24
<210> 318
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 318
Ile Leu Tyr Asp Gly Ser Ser Glu
1 5
<210> 319
<211> 36
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 319
gcgagagatt tattggcaat tggctggttc gaccgc 36
<210> 320
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 320
Ala Arg Asp Leu Leu Ala Ile Gly Trp Phe Asp Arg
1 5 10
<210> 321
<211> 324
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 321
gaaattgtgt tgacgcagtc tccaggcacc ctgactttgt ctccagggga aagagccacc 60
ctctcctgta gggccagtca gagtcttagt agttataact tagcctggta ccagcagaag 120
cctggccagg ctcccaggct cctcatctat ggtacatcca gcagggccac tggcatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag cagacttgag 240
cctgaagatt ttacagtgta ttattgtcag caatatggta gctcacctct cactttcggc 300
ggagggacca cggtggagat caaa 324
<210> 322
<211> 108
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 322
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Thr Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Leu Ser Ser Tyr
20 25 30
Asn Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Thr Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Thr Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Leu Thr Phe Gly Gly Gly Thr Thr Val Glu Ile Lys
100 105
<210> 323
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 323
cagagtctta gtagttataa c 21
<210> 324
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 324
Gln Ser Leu Ser Ser Tyr Asn
1 5
<210> 325
<211> 9
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 325
ggtacatcc 9
<210> 326
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 326
Gly Thr Ser
1
<210> 327
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 327
cagcaatatg gtagctcacc tctcact 27
<210> 328
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 328
Gln Gln Tyr Gly Ser Ser Pro Leu Thr
1 5
<210> 329
<211> 357
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 329
caggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cgtctggatt caccttcagg agttatggca tacactgggt ccgccaggct 120
ccaggcaagg gactggagtg ggtggcactt atattatatg atggaagtag tgaggactat 180
gcagactccg tgaagggccg attcaccatc tccagagaca attccaagaa tatggtgttt 240
ctgcaaatga acagcctgag agtcgaggac acggctgtct attactgtgc gagagattta 300
ttggcaattg gctggttcga ccgctggggc cagggaaccc tggtcaccgt ctcctca 357
<210> 330
<211> 119
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 330
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Arg Ser Tyr
20 25 30
Gly Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Leu Ile Leu Tyr Asp Gly Ser Ser Glu Asp Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Met Val Phe
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Val Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Leu Leu Ala Ile Gly Trp Phe Asp Arg Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 331
<211> 324
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 331
gaaattgtgt tgacgcagtc tccaggcacc ctgactttgt ctccagggga aagagccacc 60
ctctcctgta gggccagtca gagtcttagt agttataact tagcctggta ccagcagaag 120
cctggccagg ctcccaggct cctcatctat ggtacatcca gcagggccac tggcatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag cagacttgag 240
cctgaagatt ttacagtgta ttattgtcag caatatggta gctcacctct cactttcggc 300
ggagggacca aggtggagat caaa 324
<210> 332
<211> 108
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 332
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Thr Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Leu Ser Ser Tyr
20 25 30
Asn Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Thr Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Thr Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 333
<211> 357
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 333
caggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagg agttatggca tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtggcagtt atattatatg atggaagtag tgagtactat 180
gcagactccg tgaagggccg attcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agctgaggac acggctgtgt attactgtgc gagagattta 300
ttggcaattg gctggttcga ccgctggggc cagggaaccc tggtcaccgt ctcctca 357
<210> 334
<211> 119
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 334
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Arg Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Leu Tyr Asp Gly Ser Ser Glu Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Leu Leu Ala Ile Gly Trp Phe Asp Arg Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 335
<211> 325
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 335
gaaattgtgt tgacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtcttagt agttataact tagcctggta ccagcagaaa 120
cctggccagg ctcccaggct cctcatctat ggtacatcca gcagggccac tggcatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag cagactggag 240
cctgaagatt ttgcagtgta ttactgtcag caatatggta gctcacctct cactttcggc 300
ggagggacca aggtggagat caaac 325
<210> 336
<211> 108
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 336
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Leu Ser Ser Tyr
20 25 30
Asn Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Thr Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 337
<211> 354
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 337
gaggtgcagc tggtggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt acctacgaca tgaactgggt ccgccaagct 120
acaggaaaag gtctggaatg ggtctcaggt attgatactg ctggtgacac atactatcca 180
gactccgtga agggccgttt caccatctcc agagaaaatg ccaagaactc cttgtatctt 240
caaatgaaca gcctgagagt cggggacacg gctgtgtatt actgtgcaag agggggcgat 300
ttttggagtg gtccagacta ctggggccag ggaaccctgg tcaccgtctc ctca 354
<210> 338
<211> 118
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 338
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Tyr
20 25 30
Asp Met Asn Trp Val Arg Gln Ala Thr Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Asp Thr Ala Gly Asp Thr Tyr Tyr Pro Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Val Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Gly Asp Phe Trp Ser Gly Pro Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 339
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 339
ggattcacct tcagtaccta cgac 24
<210> 340
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 340
Gly Phe Thr Phe Ser Thr Tyr Asp
1 5
<210> 341
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 341
attgatactg ctggtgacac a 21
<210> 342
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 342
Ile Asp Thr Ala Gly Asp Thr
1 5
<210> 343
<211> 36
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 343
gcaagagggg gcgatttttg gagtggtcca gactac 36
<210> 344
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 344
Ala Arg Gly Gly Asp Phe Trp Ser Gly Pro Asp Tyr
1 5 10
<210> 345
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 345
gacatccaga tgacccagtc tccatcatca ctgtctgcat ctgtcggaga cagagtcacc 60
atcacttgtc gggcgagtca ggacattagc aattatttag cctggtttca gcagaaacca 120
gggaaagccc ctaagtccct gatctatgct gcattcagtt tgcaaagtgg ggtcccatca 180
aagttcagcg gcagtgtatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ttgccaacag tatattactt acccattcac tttcggccct 300
gggaccaaag tggatatcaa a 321
<210> 346
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 346
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Ala Trp Phe Gln Gln Lys Pro Gly Lys Ala Pro Lys Ser Leu Ile
35 40 45
Tyr Ala Ala Phe Ser Leu Gln Ser Gly Val Pro Ser Lys Phe Ser Gly
50 55 60
Ser Val Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Ile Thr Tyr Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 347
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 347
caggacatta gcaattat 18
<210> 348
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 348
Gln Asp Ile Ser Asn Tyr
1 5
<210> 349
<211> 9
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 349
gctgcattc 9
<210> 350
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 350
Ala Ala Phe
1
<210> 351
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 351
caacagtata ttacttaccc attcact 27
<210> 352
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 352
Gln Gln Tyr Ile Thr Tyr Pro Phe Thr
1 5
<210> 353
<211> 354
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 353
gaggtgcagc tggtggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt acctacgaca tgaactgggt ccgccaagct 120
acaggaaaag gtctggaatg ggtctcaggt attgatactg ctggtgacac atactatcca 180
gactccgtga agggccgttt caccatctcc agagaaaatg ccaagaactc cttgtatctt 240
caaatgaaca gcctgagagt cggggacacg gctgtgtatt actgtgcaag agggggcgat 300
ttttggagtg gtccagacta ctggggccag ggaaccctgg tcaccgtctc ctca 354
<210> 354
<211> 118
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 354
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Tyr
20 25 30
Asp Met Asn Trp Val Arg Gln Ala Thr Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Asp Thr Ala Gly Asp Thr Tyr Tyr Pro Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Val Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Gly Asp Phe Trp Ser Gly Pro Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 355
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 355
gacatccaga tgacccagtc tccatcatca ctgtctgcat ctgtcggaga cagagtcacc 60
atcacttgtc gggcgagtca ggacattagc aattatttag cctggtttca gcagaaacca 120
gggaaagccc ctaagtccct gatctatgct gcattcagtt tgcaaagtgg ggtcccatca 180
aagttcagcg gcagtgtatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ttgccaacag tatattactt acccattcac tttcggccct 300
gggaccaaag tggatatcaa a 321
<210> 356
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 356
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Ala Trp Phe Gln Gln Lys Pro Gly Lys Ala Pro Lys Ser Leu Ile
35 40 45
Tyr Ala Ala Phe Ser Leu Gln Ser Gly Val Pro Ser Lys Phe Ser Gly
50 55 60
Ser Val Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Ile Thr Tyr Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 357
<211> 354
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 357
gaggtgcagc tggtggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt acctacgaca tgcactgggt ccgccaagct 120
acaggaaaag gtctggagtg ggtctcagct attgatactg ctggtgacac atactatcca 180
ggctccgtga agggccgatt caccatctcc agagaaaatg ccaagaactc cttgtatctt 240
caaatgaaca gcctgagagc cggggacacg gctgtgtatt actgtgcaag agggggcgat 300
ttttggagtg gtccagacta ctggggccag ggaaccctgg tcaccgtctc ctca 354
<210> 358
<211> 118
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 358
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Tyr
20 25 30
Asp Met His Trp Val Arg Gln Ala Thr Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Asp Thr Ala Gly Asp Thr Tyr Tyr Pro Gly Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Gly Asp Phe Trp Ser Gly Pro Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 359
<211> 322
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 359
gacatccaga tgacccagtc tccatcctca ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgtc gggcgagtca ggacattagc aattatttag cctggtttca gcagaaacca 120
gggaaagccc ctaagtccct gatctatgct gcattcagtt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgccaacag tatattactt acccattcac tttcggccct 300
gggaccaaag tggatatcaa ac 322
<210> 360
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 360
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Ala Trp Phe Gln Gln Lys Pro Gly Lys Ala Pro Lys Ser Leu Ile
35 40 45
Tyr Ala Ala Phe Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Ile Thr Tyr Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 361
<211> 363
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 361
gaagtggaac tggtggagtc tgggggaggc ttggtacagc ctggcaggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttgat gattatgcca tgcactgggt ccggcaagct 120
ccagggaagg gcctggagtg ggtctcaggt attagttgga atagtggtag catagaatat 180
gcggactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctccctgtat 240
ctgcaaatga acagtctgag agctgaggac acggccttgt attactgtgc aaaagatcac 300
tgggactacg actttgaata cttccaccac tggggccagg gcaccctggt caccgtctcc 360
tca 363
<210> 362
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 362
Glu Val Glu Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Asn Ser Gly Ser Ile Glu Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Lys Asp His Trp Asp Tyr Asp Phe Glu Tyr Phe His His Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 363
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 363
ggattcacct ttgatgatta tgcc 24
<210> 364
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 364
Gly Phe Thr Phe Asp Asp Tyr Ala
1 5
<210> 365
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 365
attagttgga atagtggtag cata 24
<210> 366
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 366
Ile Ser Trp Asn Ser Gly Ser Ile
1 5
<210> 367
<211> 42
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 367
gcaaaagatc actgggacta cgactttgaa tacttccacc ac 42
<210> 368
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 368
Ala Lys Asp His Trp Asp Tyr Asp Phe Glu Tyr Phe His His
1 5 10
<210> 369
<211> 318
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 369
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt agctggttgg cctggtatca gcagaaacca 120
gggaaggccc ctaacctcct gatctataag gcgtctagtt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag tataatagtt attacacttt tggccagggg 300
accaagctgg agatcaaa 318
<210> 370
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 370
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Asn Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asn Ser Tyr Tyr Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 371
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 371
cagagtatta gtagctgg 18
<210> 372
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 372
Gln Ser Ile Ser Ser Trp
1 5
<210> 373
<211> 9
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 373
aaggcgtct 9
<210> 374
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 374
Lys Ala Ser
1
<210> 375
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 375
caacagtata atagttatta cact 24
<210> 376
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 376
Gln Gln Tyr Asn Ser Tyr Tyr Thr
1 5
<210> 377
<211> 363
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 377
gaagtgcagc tggtggagtc tgggggaggc ttggtacagc ctggcaggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttgat gattatgcca tgcactgggt ccggcaagct 120
ccagggaagg gcctggagtg ggtctcaggt attagttgga atagtggtag catagaatat 180
gcggactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctccctgtat 240
ctgcaaatga acagtctgag agctgaggac acggccttgt attactgtgc aaaagatcac 300
tgggactacg actttgaata cttccaccac tggggccagg gcaccctggt caccgtctcc 360
tca 363
<210> 378
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 378
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Asn Ser Gly Ser Ile Glu Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Lys Asp His Trp Asp Tyr Asp Phe Glu Tyr Phe His His Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 379
<211> 318
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 379
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt agctggttgg cctggtatca gcagaaacca 120
gggaaggccc ctaacctcct gatctataag gcgtctagtt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag tataatagtt attacacttt tggccagggg 300
accaagctgg agatcaaa 318
<210> 380
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 380
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Asn Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asn Ser Tyr Tyr Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 381
<211> 363
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 381
gaagtgcagc tggtggagtc tgggggaggc ttggtacagc ctggcaggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttgat gattatgcca tgcactgggt ccggcaagct 120
ccagggaagg gcctggagtg ggtctcaggt attagttgga atagtggtag cataggctat 180
gcggactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctccctgtat 240
ctgcaaatga acagtctgag agctgaggac acggccttgt attactgtgc aaaagatcac 300
tgggactacg actttgaata cttccaccac tggggccagg gcaccctggt caccgtctcc 360
tca 363
<210> 382
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 382
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Asn Ser Gly Ser Ile Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Lys Asp His Trp Asp Tyr Asp Phe Glu Tyr Phe His His Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 383
<211> 319
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 383
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt agctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctataag gcgtctagtt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag tataatagtt attacacttt tggccagggg 300
accaagctgg agatcaaac 319
<210> 384
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 384
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asn Ser Tyr Tyr Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 385
<211> 363
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 385
gaagtgcagc tggtggagtc tgggggaggc ttggtacagc ctggcaggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttgat gattatgcca tgcactgggt ccggcaagct 120
ccagggaagg gcctggagtg ggtctcaggt attagttgga atagtggtag catagtctat 180
gcggactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctccctgtat 240
ctgcaaatga acagtctaat atctgaggac acggccttgt attactgtgc aaaagatgaa 300
aacagctcgt cggggaactg gttcgacccc tggggccagg gaaccctggt caccgtctcc 360
tca 363
<210> 386
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 386
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Asn Ser Gly Ser Ile Val Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Ile Ser Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Lys Asp Glu Asn Ser Ser Ser Gly Asn Trp Phe Asp Pro Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 387
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 387
ggattcacct ttgatgatta tgcc 24
<210> 388
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 388
Gly Phe Thr Phe Asp Asp Tyr Ala
1 5
<210> 389
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 389
attagttgga atagtggtag cata 24
<210> 390
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 390
Ile Ser Trp Asn Ser Gly Ser Ile
1 5
<210> 391
<211> 42
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 391
gcaaaagatg aaaacagctc gtcggggaac tggttcgacc cc 42
<210> 392
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 392
Ala Lys Asp Glu Asn Ser Ser Ser Gly Asn Trp Phe Asp Pro
1 5 10
<210> 393
<211> 318
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 393
gaagtagtga tgacgcagtc tccagccacc ctgtctgtgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agcaacttag cctggtacca gcagaaacct 120
ggccaggctc ccaggctcct catctatggt gcatccacca gggccactgg tatcccagcc 180
aggttcagtg gcagtgggtc tgggacagag ttcactctca ccatcagcag cctgcagtct 240
gaagattttg cagtttatta ctgtcaggaa tataataatt ggatcacctt cggccaaggg 300
acacgactgg agattaaa 318
<210> 394
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 394
Glu Val Val Met Thr Gln Ser Pro Ala Thr Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Thr Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Ser
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Glu Tyr Asn Asn Trp Ile Thr
85 90 95
Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 395
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 395
cagagtgtta gcagcaac 18
<210> 396
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 396
Gln Ser Val Ser Ser Asn
1 5
<210> 397
<211> 9
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 397
ggtgcatcc 9
<210> 398
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 398
Gly Ala Ser
1
<210> 399
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 399
caggaatata ataattggat cacc 24
<210> 400
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 400
Gln Glu Tyr Asn Asn Trp Ile Thr
1 5
<210> 401
<211> 363
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 401
gaagtgcagc tggtggagtc tgggggaggc ttggtacagc ctggcaggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttgat gattatgcca tgcactgggt ccggcaagct 120
ccagggaagg gcctggagtg ggtctcaggt attagttgga atagtggtag catagtctat 180
gcggactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctccctgtat 240
ctgcaaatga acagtctaat atctgaggac acggccttgt attactgtgc aaaagatgaa 300
aacagctcgt cggggaactg gttcgacccc tggggccagg gaaccctggt caccgtctcc 360
tca 363
<210> 402
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 402
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Asn Ser Gly Ser Ile Val Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Ile Ser Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Lys Asp Glu Asn Ser Ser Ser Gly Asn Trp Phe Asp Pro Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 403
<211> 318
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 403
gaaatagtga tgacgcagtc tccagccacc ctgtctgtgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agcaacttag cctggtacca gcagaaacct 120
ggccaggctc ccaggctcct catctatggt gcatccacca gggccactgg tatcccagcc 180
aggttcagtg gcagtgggtc tgggacagag ttcactctca ccatcagcag cctgcagtct 240
gaagattttg cagtttatta ctgtcaggaa tataataatt ggatcacctt cggccaaggg 300
acacgactgg agattaaa 318
<210> 404
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 404
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Thr Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Ser
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Glu Tyr Asn Asn Trp Ile Thr
85 90 95
Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 405
<211> 363
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 405
gaagtgcagc tggtggagtc tgggggaggc ttggtacagc ctggcaggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttgat gattatgcca tgcactgggt ccggcaagct 120
ccagggaagg gcctggagtg ggtctcaggt attagttgga atagtggtag cataggctat 180
gcggactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctccctgtat 240
ctgcaaatga acagtctgag agctgaggac acggccttgt attactgtgc aaaagatgaa 300
aacagctcgt cggggaactg gttcgacccc tggggccagg gaaccctggt caccgtctcc 360
tca 363
<210> 406
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 406
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Asn Ser Gly Ser Ile Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Lys Asp Glu Asn Ser Ser Ser Gly Asn Trp Phe Asp Pro Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 407
<211> 319
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 407
gaaatagtga tgacgcagtc tccagccacc ctgtctgtgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agcaacttag cctggtacca gcagaaacct 120
ggccaggctc ccaggctcct catctatggt gcatccacca gggccactgg tatcccagcc 180
aggttcagtg gcagtgggtc tgggacagag ttcactctca ccatcagcag cctgcagtct 240
gaagattttg cagtttatta ctgtcaggaa tataataatt ggatcacctt cggccaaggg 300
acacgactgg agattaaac 319
<210> 408
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 408
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Thr Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Ser
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Glu Tyr Asn Asn Trp Ile Thr
85 90 95
Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 409
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 409
gaggtgcagc tggtggagtc tgggggaggc ttggtaaagc ctggggggtc ccttagactc 60
tcctgtgcag cctctggatt cactttcagt aacgcctgga tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggttggccat attcaaagca aaactgatgg tgggacaaca 180
gactacgctg cacccgtgaa aggcagattc accatctcaa gagatgattc aaaaaacacg 240
ctgtatctgc aaatgaacag cctgaaaacc gaggacacag ccgtgtatta ctgtacaaca 300
gcagattacg atttttggag tggggttgac tactggggcc agggaaccct ggtcaccgtc 360
tcttca 366
<210> 410
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 410
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly His Ile Gln Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Ala Asp Tyr Asp Phe Trp Ser Gly Val Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 411
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 411
ggattcactt tcagtaacgc ctgg 24
<210> 412
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 412
Gly Phe Thr Phe Ser Asn Ala Trp
1 5
<210> 413
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 413
attcaaagca aaactgatgg tgggacaaca 30
<210> 414
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 414
Ile Gln Ser Lys Thr Asp Gly Gly Thr Thr
1 5 10
<210> 415
<211> 39
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 415
acaacagcag attacgattt ttggagtggg gttgactac 39
<210> 416
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 416
Thr Thr Ala Asp Tyr Asp Phe Trp Ser Gly Val Asp Tyr
1 5 10
<210> 417
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 417
gacatccaga tgacccagtc tccatcttcc gtgtctgcat ctgtaggaga cagagtcacc 60
atcacttgtc gggcgagtca gggtattagc agctggttag tctggtatca gcagaaacca 120
gggaaggccc ctaagctcct gatttatgct gcatccagtt tacaaagtgg ggtcccatca 180
agattcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttacta ttgtcaacag gctaacagtt tcccattcac tttcggccct 300
gggaccaaag tggatgtcaa a 321
<210> 418
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 418
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Ser Trp
20 25 30
Leu Val Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Asn Ser Phe Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Val Lys
100 105
<210> 419
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 419
cagggtatta gcagctgg 18
<210> 420
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 420
Gln Gly Ile Ser Ser Trp
1 5
<210> 421
<211> 9
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 421
gctgcatcc 9
<210> 422
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 422
Ala Ala Ser
1
<210> 423
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 423
caacaggcta acagtttccc attcact 27
<210> 424
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 424
Gln Gln Ala Asn Ser Phe Pro Phe Thr
1 5
<210> 425
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 425
gaggtgcagc tggtggagtc tgggggaggc ttggtaaagc ctggggggtc ccttagactc 60
tcctgtgcag cctctggatt cactttcagt aacgcctgga tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggttggccat attcaaagca aaactgatgg tgggacaaca 180
gactacgctg cacccgtgaa aggcagattc accatctcaa gagatgattc aaaaaacacg 240
ctgtatctgc aaatgaacag cctgaaaacc gaggacacag ccgtgtatta ctgtacaaca 300
gcagattacg atttttggag tggggttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 426
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 426
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly His Ile Gln Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Ala Asp Tyr Asp Phe Trp Ser Gly Val Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 427
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 427
gacatccaga tgacccagtc tccatcttcc gtgtctgcat ctgtaggaga cagagtcacc 60
atcacttgtc gggcgagtca gggtattagc agctggttag tctggtatca gcagaaacca 120
gggaaggccc ctaagctcct gatttatgct gcatccagtt tacaaagtgg ggtcccatca 180
agattcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttacta ttgtcaacag gctaacagtt tcccattcac tttcggccct 300
gggaccaaag tggatatcaa a 321
<210> 428
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 428
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Ser Trp
20 25 30
Leu Val Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Asn Ser Phe Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 429
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 429
gaggtgcagc tggtggagtc tgggggaggc ttggtaaagc ctggggggtc ccttagactc 60
tcctgtgcag cctctggatt cactttcagt aacgcctgga tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggttggccgt attcaaagca aaactgatgg tgggacaaca 180
gactacgctg cacccgtgaa aggcagattc accatctcaa gagatgattc aaaaaacacg 240
ctgtatctgc aaatgaacag cctgaaaacc gaggacacag ccgtgtatta ctgtacaaca 300
gcagattacg atttttggag tggggttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 430
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 430
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Gln Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Ala Asp Tyr Asp Phe Trp Ser Gly Val Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 431
<211> 322
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 431
gacatccaga tgacccagtc tccatcttct gtgtctgcat ctgtaggaga cagagtcacc 60
atcacttgtc gggcgagtca gggtattagc agctggttag cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ctatcagcag cctgcagcct 240
gaagattttg caacttacta ttgtcaacag gctaacagtt tcccattcac tttcggccct 300
gggaccaaag tggatatcaa ac 322
<210> 432
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 432
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Asn Ser Phe Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 433
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 433
gaggtgcagc tggtggagtc tgggggaggc ttggtaaagc ctggggggtc ccttagactc 60
tcctgtgcag cctctggatt cactttcagt aacgcctgga tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggttggccgt attaaaagca aaactgatgg tgggacaaca 180
gactacgctg cacccgtgaa aggcagattc accatctcaa gagatgattc aaaaaacacg 240
ctgtatctgc aaatgaacag cctgaaaacc gaggacacag ccgtgtatta ctgtatcaca 300
gcagattacg atttttggag tggggttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 434
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 434
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ile Thr Ala Asp Tyr Asp Phe Trp Ser Gly Val Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 435
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 435
ggattcactt tcagtaacgc ctgg 24
<210> 436
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 436
Gly Phe Thr Phe Ser Asn Ala Trp
1 5
<210> 437
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 437
attaaaagca aaactgatgg tgggacaaca 30
<210> 438
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 438
Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr
1 5 10
<210> 439
<211> 39
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 439
atcacagcag attacgattt ttggagtggg gttgactac 39
<210> 440
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 440
Ile Thr Ala Asp Tyr Asp Phe Trp Ser Gly Val Asp Tyr
1 5 10
<210> 441
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 441
gacatccaga tgacccagtc tccatcttcc gtgtctgcat ctgtaggaga cagagtcacc 60
atcacttgtc gggcgagtca gggtattagc agctggttag cctggtatca gcagaaacca 120
gggaaagccc ctaaactcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttacta ttgtcaacag gctaacagtt tcccattcac tttcggccct 300
gggaccaaag tggatatcaa a 321
<210> 442
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 442
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Asn Ser Phe Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 443
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 443
cagggtatta gcagctgg 18
<210> 444
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 444
Gln Gly Ile Ser Ser Trp
1 5
<210> 445
<211> 9
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 445
gctgcatcc 9
<210> 446
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 446
Ala Ala Ser
1
<210> 447
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 447
caacaggcta acagtttccc attcact 27
<210> 448
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 448
Gln Gln Ala Asn Ser Phe Pro Phe Thr
1 5
<210> 449
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 449
gaggtgcagc tggtggagtc tgggggaggc ttggtaaagc ctggggggtc ccttagactc 60
tcctgtgcag cctctggatt cactttcagt aacgcctgga tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggttggccgt attaaaagca aaactgatgg tgggacaaca 180
gactacgctg cacccgtgaa aggcagattc accatctcaa gagatgattc aaaaaacacg 240
ctgtatctgc aaatgaacag cctgaaaacc gaggacacag ccgtgtatta ctgtatcaca 300
gcagattacg atttttggag tggggttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 450
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 450
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ile Thr Ala Asp Tyr Asp Phe Trp Ser Gly Val Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 451
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 451
gacatccaga tgacccagtc tccatcttcc gtgtctgcat ctgtaggaga cagagtcacc 60
atcacttgtc gggcgagtca gggtattagc agctggttag cctggtatca gcagaaacca 120
gggaaagccc ctaaactcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttacta ttgtcaacag gctaacagtt tcccattcac tttcggccct 300
gggaccaaag tggatatcaa a 321
<210> 452
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 452
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Asn Ser Phe Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 453
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 453
gaggtgcagc tggtggagtc tgggggaggc ttggtaaagc ctggggggtc ccttagactc 60
tcctgtgcag cctctggatt cactttcagt aacgcctgga tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggttggccgt attaaaagca aaactgatgg tgggacaaca 180
gactacgctg cacccgtgaa aggcagattc accatctcaa gagatgattc aaaaaacacg 240
ctgtatctgc aaatgaacag cctgaaaacc gaggacacag ccgtgtatta ctgtatcaca 300
gcagattacg atttttggag tggggttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 454
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 454
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ile Thr Ala Asp Tyr Asp Phe Trp Ser Gly Val Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 455
<211> 322
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 455
gacatccaga tgacccagtc tccatcttct gtgtctgcat ctgtaggaga cagagtcacc 60
atcacttgtc gggcgagtca gggtattagc agctggttag cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ctatcagcag cctgcagcct 240
gaagattttg caacttacta ttgtcaacag gctaacagtt tcccattcac tttcggccct 300
gggaccaaag tggatatcaa ac 322
<210> 456
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 456
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Asn Ser Phe Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 457
<211> 363
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 457
gaagtgcagc tggtggagtc tgggggaggc ttggtacagc ctggcaggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttgat gattatgcca tgcactgggt ccggcaagct 120
ccagggaagg gcctggagtg ggtctcaggt attagttgga atagtggtag catagtctat 180
gcggactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctccctgtat 240
ctgcaaatga acagtctaat atctgaggac acggccttgt attactgtgc aaaagatgaa 300
agcagctcgt cggggaactg gttcgacccc tggggccagg gaaccctggt caccgtctcc 360
tca 363
<210> 458
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 458
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Asn Ser Gly Ser Ile Val Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Ile Ser Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Lys Asp Glu Ser Ser Ser Ser Gly Asn Trp Phe Asp Pro Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 459
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 459
ggattcacct ttgatgatta tgcc 24
<210> 460
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 460
Gly Phe Thr Phe Asp Asp Tyr Ala
1 5
<210> 461
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 461
attagttgga atagtggtag cata 24
<210> 462
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 462
Ile Ser Trp Asn Ser Gly Ser Ile
1 5
<210> 463
<211> 42
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 463
gcaaaagatg aaagcagctc gtcggggaac tggttcgacc cc 42
<210> 464
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 464
Ala Lys Asp Glu Ser Ser Ser Ser Gly Asn Trp Phe Asp Pro
1 5 10
<210> 465
<211> 363
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 465
gaagtgcagc tggtggagtc tgggggaggc ttggtacagc ctggcaggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttgat gattatgcca tgcactgggt ccggcaagct 120
ccagggaagg gcctggagtg ggtctcaggt attagttgga atagtggtag catagtctat 180
gcggactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctccctgtat 240
ctgcaaatga acagtctaat atctgaggac acggccttgt attactgtgc aaaagatgaa 300
agcagctcgt cggggaactg gttcgacccc tggggccagg gaaccctggt caccgtctcc 360
tca 363
<210> 466
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 466
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Asn Ser Gly Ser Ile Val Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Ile Ser Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Lys Asp Glu Ser Ser Ser Ser Gly Asn Trp Phe Asp Pro Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 467
<211> 363
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 467
gaagtgcagc tggtggagtc tgggggaggc ttggtacagc ctggcaggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttgat gattatgcca tgcactgggt ccggcaagct 120
ccagggaagg gcctggagtg ggtctcaggt attagttgga atagtggtag cataggctat 180
gcggactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctccctgtat 240
ctgcaaatga acagtctgag agctgaggac acggccttgt attactgtgc aaaagatgaa 300
agcagctcgt cggggaactg gttcgacccc tggggccagg gaaccctggt caccgtctcc 360
tca 363
<210> 468
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 468
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Asn Ser Gly Ser Ile Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Lys Asp Glu Ser Ser Ser Ser Gly Asn Trp Phe Asp Pro Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 469
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<220>
<221> VARIANT
<222> (1)...(1)
<223> Xaa = Gly
<220>
<221> VARIANT
<222> (2)...(2)
<223> Xaa = Gly or Phe
<220>
<221> VARIANT
<222> (3)...(3)
<223> Xaa = Ser or Thr
<220>
<221> VARIANT
<222> (4)...(4)
<223> Xaa = Phe
<220>
<221> VARIANT
<222> (5)...(5)
<223> Xaa = Ser
<220>
<221> VARIANT
<222> (6)...(6)
<223> Xaa = Ile, Ser, or Thr
<220>
<221> VARIANT
<222> (7)...(7)
<223> Xaa = His or Tyr
<220>
<221> VARIANT
<222> (8)...(8)
<223> Xaa = His, Gly, or Asp
<400> 469
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5
<210> 470
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<220>
<221> VARIANT
<222> (1)...(1)
<223> Xaa = Ile
<220>
<221> VARIANT
<222> (2)...(2)
<223> Xaa = Asn, Ser, or Gly
<220>
<221> VARIANT
<222> (3)...(3)
<223> Xaa = His, Phe, Ser, or Val
<220>
<221> VARIANT
<222> (4)...(4)
<223> Xaa = Arg, Asp, or Ala
<220>
<221> VARIANT
<222> (5)...(5)
<223> Xaa = Gly
<220>
<221> VARIANT
<222> (6)...(6)
<223> Xaa = Gly or absent
<220>
<221> VARIANT
<222> (7)...(7)
<223> Xaa = Ser, Asn, or Asp
<220>
<221> VARIANT
<222> (8)...(8)
<223> Xaa = Thr or Lys
<400> 470
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5
<210> 471
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<220>
<221> VARIANT
<222> (1)...(1)
<223> Xaa = Ala
<220>
<221> VARIANT
<222> (2)...(2)
<223> Xaa = Arg or Lys
<220>
<221> VARIANT
<222> (3)...(3)
<223> Xaa = Gly or Glu
<220>
<221> VARIANT
<222> (4)...(4)
<223> Xaa = Gly, Asp, or absent
<220>
<221> VARIANT
<222> (5)...(5)
<223> Xaa = Asp or absent
<220>
<221> VARIANT
<222> (6)...(6)
<223> Xaa = Leu, Arg, or absent
<220>
<221> VARIANT
<222> (7)...(7)
<223> Xaa = Arg or Ser
<220>
<221> VARIANT
<222> (8)...(8)
<223> Xaa = Phe, Gly, or Arg
<220>
<221> VARIANT
<222> (9)...(9)
<223> Xaa = Leu, His, or Asn
<220>
<221> VARIANT
<222> (10)...(10)
<223> Xaa = Asp, Pro, or Tyr
<220>
<221> VARIANT
<222> (11)...(11)
<223> Xaa = Trp, Tyr, or Phe
<220>
<221> VARIANT
<222> (12)...(12)
<223> Xaa = Leu, Phe, Val, or Asp
<220>
<221> VARIANT
<222> (13)...(13)
<223> Xaa = Ser, Tyr, or Gly
<220>
<221> VARIANT
<222> (14)...(14)
<223> Xaa = Ser, Tyr, or Asp
<220>
<221> VARIANT
<222> (15)...(15)
<223> Xaa = Tyr
<220>
<221> VARIANT
<222> (16)...(16)
<223> Xaa = Phe or Gly
<220>
<221> VARIANT
<222> (17)...(17)
<223> Xaa = Leu or absent
<220>
<221> VARIANT
<222> (18)...(18)
<223> Xaa = Asp
<220>
<221> VARIANT
<222> (19)...(19)
<223> Xaa = Tyr, Val, or Phe
<220>
<221> VARIANT
<222> (20)...(20)
<223> Xaa = Trp
<400> 471
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10 15
Xaa Xaa Xaa Xaa
20
<210> 472
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<220>
<221> VARIANT
<222> (1)...(1)
<223> Xaa = Gln
<220>
<221> VARIANT
<222> (2)...(2)
<223> Xaa = Gly or Ser
<220>
<221> VARIANT
<222> (3)...(3)
<223> Xaa = Ile
<220>
<221> VARIANT
<222> (4)...(4)
<223> Xaa = Ser or Asn
<220>
<221> VARIANT
<222> (5)...(5)
<223> Xaa = Asp, Ser, or Arg
<220>
<221> VARIANT
<222> (6)...(6)
<223> Xaa = Tyr or Trp
<400> 472
Xaa Xaa Xaa Xaa Xaa Xaa
1 5
<210> 473
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<220>
<221> VARIANT
<222> (1)...(1)
<223> Xaa = Ala or Lys
<220>
<221> VARIANT
<222> (2)...(2)
<223> Xaa = Ala
<220>
<221> VARIANT
<222> (3)...(3)
<223> Xaa = Ser
<400> 473
Xaa Xaa Xaa
1
<210> 474
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<220>
<221> VARIANT
<222> (1)...(1)
<223> Xaa = Gln
<220>
<221> VARIANT
<222> (2)...(2)
<223> Xaa = Asn or Gln
<220>
<221> VARIANT
<222> (3)...(3)
<223> Xaa = Tyr or Leu
<220>
<221> VARIANT
<222> (4)...(4)
<223> Xaa = Asn, His, Ser, or Asp
<220>
<221> VARIANT
<222> (5)...(5)
<223> Xaa = Thr or Ser
<220>
<221> VARIANT
<222> (6)...(6)
<223> Xaa = Ala or Tyr
<220>
<221> VARIANT
<222> (7)...(7)
<223> Xaa = Pro, Ser, or Phe
<220>
<221> VARIANT
<222> (8)...(8)
<223> Xaa = Leu or Arg
<220>
<221> VARIANT
<222> (9)...(9)
<223> Xaa = Thr
<220>
<221> VARIANT
<222> (10)...(10)
<223> Xaa = Phe
<400> 474
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10
<210> 475
<211> 1221
<212> ДНК
<213> Homo sapiens
<400> 475
atgagcggtg ctccgacggc cggggcagcc ctgatgctct gcgccgccac cgccgtgcta 60
ctgagcgctc agggcggacc cgtgcagtcc aagtcgccgc gctttgcgtc ctgggacgag 120
atgaatgtcc tggcgcacgg actcctgcag ctcggccagg ggctgcgcga acacgcggag 180
cgcacccgca gtcagctgag cgcgctggag cggcgcctga gcgcgtgcgg gtccgcctgt 240
cagggaaccg aggggtccac cgacctcccg ttagcccctg agagccgggt ggaccctgag 300
gtccttcaca gcctgcagac acaactcaag gctcagaaca gcaggatcca gcaactcttc 360
cacaaggtgg cccagcagca gcggcacctg gagaagcagc acctgcgaat tcagcatctg 420
caaagccagt ttggcctcct ggaccacaag cacctagacc atgaggtggc caagcctgcc 480
cgaagaaaga ggctgcccga gatggcccag ccagttgacc cggctcacaa tgtcagccgc 540
ctgcaccggc tgcccaggga ttgccaggag ctgttccagg ttggggagag gcagagtgga 600
ctatttgaaa tccagcctca ggggtctccg ccatttttgg tgaactgcaa gatgacctca 660
gatggaggct ggacagtaat tcagaggcgc cacgatggct cagtggactt caaccggccc 720
tgggaagcct acaaggcggg gtttggggat ccccacggcg agttctggct gggtctggag 780
aaggtgcata gcatcacggg ggaccgcaac agccgcctgg ccgtgcagct gcgggactgg 840
gatggcaacg ccgagttgct gcagttctcc gtgcacctgg gtggcgagga cacggcctat 900
agcctgcagc tcactgcacc cgtggccggc cagctgggcg ccaccaccgt cccacccagc 960
ggcctctccg tacccttctc cacttgggac caggatcacg acctccgcag ggacaagaac 1020
tgcgccaaga gcctctctgg aggctggtgg tttggcacct gcagccattc caacctcaac 1080
ggccagtact tccgctccat cccacagcag cggcagaagc ttaagaaggg aatcttctgg 1140
aagacctggc ggggccgcta ctacccgctg caggccacca ccatgttgat ccagcccatg 1200
gcagcagagg cagcctccta g 1221
<210> 476
<211> 406
<212> Белок
<213> Homo sapiens
<400> 476
Met Ser Gly Ala Pro Thr Ala Gly Ala Ala Leu Met Leu Cys Ala Ala
1 5 10 15
Thr Ala Val Leu Leu Ser Ala Gln Gly Gly Pro Val Gln Ser Lys Ser
20 25 30
Pro Arg Phe Ala Ser Trp Asp Glu Met Asn Val Leu Ala His Gly Leu
35 40 45
Leu Gln Leu Gly Gln Gly Leu Arg Glu His Ala Glu Arg Thr Arg Ser
50 55 60
Gln Leu Ser Ala Leu Glu Arg Arg Leu Ser Ala Cys Gly Ser Ala Cys
65 70 75 80
Gln Gly Thr Glu Gly Ser Thr Asp Leu Pro Leu Ala Pro Glu Ser Arg
85 90 95
Val Asp Pro Glu Val Leu His Ser Leu Gln Thr Gln Leu Lys Ala Gln
100 105 110
Asn Ser Arg Ile Gln Gln Leu Phe His Lys Val Ala Gln Gln Gln Arg
115 120 125
His Leu Glu Lys Gln His Leu Arg Ile Gln His Leu Gln Ser Gln Phe
130 135 140
Gly Leu Leu Asp His Lys His Leu Asp His Glu Val Ala Lys Pro Ala
145 150 155 160
Arg Arg Lys Arg Leu Pro Glu Met Ala Gln Pro Val Asp Pro Ala His
165 170 175
Asn Val Ser Arg Leu His Arg Leu Pro Arg Asp Cys Gln Glu Leu Phe
180 185 190
Gln Val Gly Glu Arg Gln Ser Gly Leu Phe Glu Ile Gln Pro Gln Gly
195 200 205
Ser Pro Pro Phe Leu Val Asn Cys Lys Met Thr Ser Asp Gly Gly Trp
210 215 220
Thr Val Ile Gln Arg Arg His Asp Gly Ser Val Asp Phe Asn Arg Pro
225 230 235 240
Trp Glu Ala Tyr Lys Ala Gly Phe Gly Asp Pro His Gly Glu Phe Trp
245 250 255
Leu Gly Leu Glu Lys Val His Ser Ile Thr Gly Asp Arg Asn Ser Arg
260 265 270
Leu Ala Val Gln Leu Arg Asp Trp Asp Gly Asn Ala Glu Leu Leu Gln
275 280 285
Phe Ser Val His Leu Gly Gly Glu Asp Thr Ala Tyr Ser Leu Gln Leu
290 295 300
Thr Ala Pro Val Ala Gly Gln Leu Gly Ala Thr Thr Val Pro Pro Ser
305 310 315 320
Gly Leu Ser Val Pro Phe Ser Thr Trp Asp Gln Asp His Asp Leu Arg
325 330 335
Arg Asp Lys Asn Cys Ala Lys Ser Leu Ser Gly Gly Trp Trp Phe Gly
340 345 350
Thr Cys Ser His Ser Asn Leu Asn Gly Gln Tyr Phe Arg Ser Ile Pro
355 360 365
Gln Gln Arg Gln Lys Leu Lys Lys Gly Ile Phe Trp Lys Thr Trp Arg
370 375 380
Gly Arg Tyr Tyr Pro Leu Gln Ala Thr Thr Met Leu Ile Gln Pro Met
385 390 395 400
Ala Ala Glu Ala Ala Ser
405
<210> 477
<211> 1233
<212> ДНК
<213> Mus muscular
<400> 477
atgcgctgcg ctccgacagc aggcgctgcc ctggtgctat gcgcggctac tgcggggctt 60
ttgagcgcgc aagggcgccc tgcacagcca gagccaccgc gctttgcatc ctgggacgag 120
atgaacttgc tggctcacgg gctgctacag ctcggccatg ggctgcgcga acacgtggag 180
cgcacccgtg ggcagctggg cgcgctggag cgccgcatgg ctgcctgtgg taacgcttgt 240
caggggccca agggaaaaga tgcacccttc aaagactccg aggatagagt ccctgaaggc 300
cagactcctg agactctgca gagtttgcag actcagctca aggctcaaaa cagcaagatc 360
cagcaattgt tccagaaggt ggcccagcag cagagatacc tatcaaagca gaatctgaga 420
atacagaatc ttcagagcca gatagacctc ttggccccca cgcacctaga caatggagta 480
gacaagactt cgaggggaaa gaggcttccc aagatgaccc agctcattgg cttgactccc 540
aacgccaccc acttacacag gccgccccgg gactgccagg aactcttcca agaaggggag 600
cggcacagtg gacttttcca gatccagcct ctggggtctc caccattttt ggtcaactgt 660
gagatgactt cagatggagg ctggacagtg attcagagac gcctgaacgg ctctgtggac 720
ttcaaccagt cctgggaagc ctacaaggat ggcttcggag atccccaagg cgagttctgg 780
ctgggcctgg aaaagatgca cagcatcaca gggaaccgag gaagccaatt ggctgtgcag 840
ctccaggact gggatggcaa tgccaaattg ctccaatttc ccatccattt ggggggtgag 900
gacacagcct acagcctgca gctcactgag cccacggcca atgagctggg tgccaccaat 960
gtttccccca atggcctttc cctgcccttc tctacttggg accaagacca tgacctccgt 1020
ggggacctta actgtgccaa gagcctctct ggtggctggt ggtttggtac ctgtagccat 1080
tccaatctca atggacaata cttccactct atcccacggc aacggcagga gcgtaaaaag 1140
ggtatcttct ggaaaacatg gaagggccgc tactatcctc tgcaggctac caccctgctg 1200
atccagccca tggaggctac agcagcctct tag 1233
<210> 478
<211> 410
<212> Белок
<213> Mus muscular
<400> 478
Met Arg Cys Ala Pro Thr Ala Gly Ala Ala Leu Val Leu Cys Ala Ala
1 5 10 15
Thr Ala Gly Leu Leu Ser Ala Gln Gly Arg Pro Ala Gln Pro Glu Pro
20 25 30
Pro Arg Phe Ala Ser Trp Asp Glu Met Asn Leu Leu Ala His Gly Leu
35 40 45
Leu Gln Leu Gly His Gly Leu Arg Glu His Val Glu Arg Thr Arg Gly
50 55 60
Gln Leu Gly Ala Leu Glu Arg Arg Met Ala Ala Cys Gly Asn Ala Cys
65 70 75 80
Gln Gly Pro Lys Gly Lys Asp Ala Pro Phe Lys Asp Ser Glu Asp Arg
85 90 95
Val Pro Glu Gly Gln Thr Pro Glu Thr Leu Gln Ser Leu Gln Thr Gln
100 105 110
Leu Lys Ala Gln Asn Ser Lys Ile Gln Gln Leu Phe Gln Lys Val Ala
115 120 125
Gln Gln Gln Arg Tyr Leu Ser Lys Gln Asn Leu Arg Ile Gln Asn Leu
130 135 140
Gln Ser Gln Ile Asp Leu Leu Ala Pro Thr His Leu Asp Asn Gly Val
145 150 155 160
Asp Lys Thr Ser Arg Gly Lys Arg Leu Pro Lys Met Thr Gln Leu Ile
165 170 175
Gly Leu Thr Pro Asn Ala Thr His Leu His Arg Pro Pro Arg Asp Cys
180 185 190
Gln Glu Leu Phe Gln Glu Gly Glu Arg His Ser Gly Leu Phe Gln Ile
195 200 205
Gln Pro Leu Gly Ser Pro Pro Phe Leu Val Asn Cys Glu Met Thr Ser
210 215 220
Asp Gly Gly Trp Thr Val Ile Gln Arg Arg Leu Asn Gly Ser Val Asp
225 230 235 240
Phe Asn Gln Ser Trp Glu Ala Tyr Lys Asp Gly Phe Gly Asp Pro Gln
245 250 255
Gly Glu Phe Trp Leu Gly Leu Glu Lys Met His Ser Ile Thr Gly Asn
260 265 270
Arg Gly Ser Gln Leu Ala Val Gln Leu Gln Asp Trp Asp Gly Asn Ala
275 280 285
Lys Leu Leu Gln Phe Pro Ile His Leu Gly Gly Glu Asp Thr Ala Tyr
290 295 300
Ser Leu Gln Leu Thr Glu Pro Thr Ala Asn Glu Leu Gly Ala Thr Asn
305 310 315 320
Val Ser Pro Asn Gly Leu Ser Leu Pro Phe Ser Thr Trp Asp Gln Asp
325 330 335
His Asp Leu Arg Gly Asp Leu Asn Cys Ala Lys Ser Leu Ser Gly Gly
340 345 350
Trp Trp Phe Gly Thr Cys Ser His Ser Asn Leu Asn Gly Gln Tyr Phe
355 360 365
His Ser Ile Pro Arg Gln Arg Gln Glu Arg Lys Lys Gly Ile Phe Trp
370 375 380
Lys Thr Trp Lys Gly Arg Tyr Tyr Pro Leu Gln Ala Thr Thr Leu Leu
385 390 395 400
Ile Gln Pro Met Glu Ala Thr Ala Ala Ser
405 410
<210> 479
<211> 1110
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 479
ggacccgtgc agtccaagtc gccgcgcttt gcgtcctggg acgagatgaa tgtcctggcg 60
cacggactcc tgcagctcgg ccaggggctg cgcgaacacg cggagcgcac ccgcagtcag 120
ctgagcgcgc tggagcggcg cctgagcgcg tgcgggtccg cctgtcaggg aaccgagggg 180
tccaccgacc tcccgttagc ccctgagagc cgggtggacc ctgaggtcct tcacagcctg 240
cagacacaac tcaaggctca gaacagcagg atccagcaac tcttccacaa ggtggcccag 300
cagcagcggc acctggagaa gcagcacctg cgaattcagc atctgcaaag ccagtttggc 360
ctcctggacc ccgggtctct cgaattcgcc cttgagagaa actccggaga gcccagaggg 420
cccacaatca agccctgtcc tccatgcaaa tgcccagcac ctaacctctt gggtggacca 480
tccgtcttca tcttccctcc aaagatcaag gatgtactca tgatctccct gagccccata 540
gtcacatgtg tggtggtgga tgtgagcgag gatgacccag atgtccagat cagctggttt 600
gtgaacaacg tggaagtaca cacagctcag acacaaaccc atagagagga ttacaacagt 660
actctccggg tggtcagtgc cctccccatc cagcaccagg actggatgag tggcaaggag 720
ttcaaatgca aggtcaacaa caaagacctc ccagcgccca tcgagagaac catctcaaaa 780
cccaaagggt cagtaagagc tccacaggta tatgtcttgc ctccaccaga agaagagatg 840
actaagaaac aggtcactct gacctgcatg gtcacagact tcatgcctga agacatttac 900
gtggagtgga ccaacaacgg gaaaacagag ctaaactaca agaacactga accagtcctg 960
gactctgatg gttcttactt catgtacagc aagctgagag tggaaaagaa gaactgggtg 1020
gaaagaaata gctactcctg ttcagtggtc cacgagggtc tgcacaatca ccacacgact 1080
aagagcttct cccggactcc gggtaaatga 1110
<210> 480
<211> 369
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 480
Gly Pro Val Gln Ser Lys Ser Pro Arg Phe Ala Ser Trp Asp Glu Met
1 5 10 15
Asn Val Leu Ala His Gly Leu Leu Gln Leu Gly Gln Gly Leu Arg Glu
20 25 30
His Ala Glu Arg Thr Arg Ser Gln Leu Ser Ala Leu Glu Arg Arg Leu
35 40 45
Ser Ala Cys Gly Ser Ala Cys Gln Gly Thr Glu Gly Ser Thr Asp Leu
50 55 60
Pro Leu Ala Pro Glu Ser Arg Val Asp Pro Glu Val Leu His Ser Leu
65 70 75 80
Gln Thr Gln Leu Lys Ala Gln Asn Ser Arg Ile Gln Gln Leu Phe His
85 90 95
Lys Val Ala Gln Gln Gln Arg His Leu Glu Lys Gln His Leu Arg Ile
100 105 110
Gln His Leu Gln Ser Gln Phe Gly Leu Leu Asp Pro Gly Ser Leu Glu
115 120 125
Phe Ala Leu Glu Arg Asn Ser Gly Glu Pro Arg Gly Pro Thr Ile Lys
130 135 140
Pro Cys Pro Pro Cys Lys Cys Pro Ala Pro Asn Leu Leu Gly Gly Pro
145 150 155 160
Ser Val Phe Ile Phe Pro Pro Lys Ile Lys Asp Val Leu Met Ile Ser
165 170 175
Leu Ser Pro Ile Val Thr Cys Val Val Val Asp Val Ser Glu Asp Asp
180 185 190
Pro Asp Val Gln Ile Ser Trp Phe Val Asn Asn Val Glu Val His Thr
195 200 205
Ala Gln Thr Gln Thr His Arg Glu Asp Tyr Asn Ser Thr Leu Arg Val
210 215 220
Val Ser Ala Leu Pro Ile Gln His Gln Asp Trp Met Ser Gly Lys Glu
225 230 235 240
Phe Lys Cys Lys Val Asn Asn Lys Asp Leu Pro Ala Pro Ile Glu Arg
245 250 255
Thr Ile Ser Lys Pro Lys Gly Ser Val Arg Ala Pro Gln Val Tyr Val
260 265 270
Leu Pro Pro Pro Glu Glu Glu Met Thr Lys Lys Gln Val Thr Leu Thr
275 280 285
Cys Met Val Thr Asp Phe Met Pro Glu Asp Ile Tyr Val Glu Trp Thr
290 295 300
Asn Asn Gly Lys Thr Glu Leu Asn Tyr Lys Asn Thr Glu Pro Val Leu
305 310 315 320
Asp Ser Asp Gly Ser Tyr Phe Met Tyr Ser Lys Leu Arg Val Glu Lys
325 330 335
Lys Asn Trp Val Glu Arg Asn Ser Tyr Ser Cys Ser Val Val His Glu
340 345 350
Gly Leu His Asn His His Thr Thr Lys Ser Phe Ser Arg Thr Pro Gly
355 360 365
Lys
<210> 481
<211> 330
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 481
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
225 230 235 240
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
290 295 300
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
305 310 315 320
Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
325 330
<210> 482
<211> 327
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 482
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Lys Thr
65 70 75 80
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro Ser Cys Pro Ala Pro
100 105 110
Glu Phe Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
115 120 125
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
130 135 140
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
145 150 155 160
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
165 170 175
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
180 185 190
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
195 200 205
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
210 215 220
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
225 230 235 240
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
245 250 255
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
260 265 270
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
275 280 285
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
290 295 300
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
305 310 315 320
Leu Ser Leu Ser Leu Gly Lys
325
<210> 483
<211> 327
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 483
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Lys Thr
65 70 75 80
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro
100 105 110
Glu Phe Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
115 120 125
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
130 135 140
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
145 150 155 160
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
165 170 175
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
180 185 190
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
195 200 205
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
210 215 220
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
225 230 235 240
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
245 250 255
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
260 265 270
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
275 280 285
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
290 295 300
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
305 310 315 320
Leu Ser Leu Ser Leu Gly Lys
325
<210> 484
<211> 455
<212> Белок
<213> Mus muscular
<400> 484
Met His Thr Ile Lys Leu Phe Leu Phe Val Val Pro Leu Val Ile Ala
1 5 10 15
Ser Arg Val Asp Pro Asp Leu Ser Ser Phe Asp Ser Ala Pro Ser Glu
20 25 30
Pro Lys Ser Arg Phe Ala Met Leu Asp Asp Val Lys Ile Leu Ala Asn
35 40 45
Gly Leu Leu Gln Leu Gly His Gly Leu Lys Asp Phe Val His Lys Thr
50 55 60
Lys Gly Gln Ile Asn Asp Ile Phe Gln Lys Leu Asn Ile Phe Asp Gln
65 70 75 80
Ser Phe Tyr Asp Leu Ser Leu Arg Thr Asn Glu Ile Lys Glu Glu Glu
85 90 95
Lys Glu Leu Arg Arg Thr Thr Ser Thr Leu Gln Val Lys Asn Glu Glu
100 105 110
Val Lys Asn Met Ser Val Glu Leu Asn Ser Lys Leu Glu Ser Leu Leu
115 120 125
Glu Glu Lys Thr Ala Leu Gln His Lys Val Arg Ala Leu Glu Glu Gln
130 135 140
Leu Thr Asn Leu Ile Leu Ser Pro Ala Gly Ala Gln Glu His Pro Glu
145 150 155 160
Val Thr Ser Leu Lys Ser Phe Val Glu Gln Gln Asp Asn Ser Ile Arg
165 170 175
Glu Leu Leu Gln Ser Val Glu Glu Gln Tyr Lys Gln Leu Ser Gln Gln
180 185 190
His Met Gln Ile Lys Glu Ile Glu Lys Gln Leu Arg Lys Thr Gly Ile
195 200 205
Gln Glu Pro Ser Glu Asn Ser Leu Ser Ser Lys Ser Arg Ala Pro Arg
210 215 220
Thr Thr Pro Pro Leu Gln Leu Asn Glu Thr Glu Asn Thr Glu Gln Asp
225 230 235 240
Asp Leu Pro Ala Asp Cys Ser Ala Val Tyr Asn Arg Gly Glu His Thr
245 250 255
Ser Gly Val Tyr Thr Ile Lys Pro Arg Asn Ser Gln Gly Phe Asn Val
260 265 270
Tyr Cys Asp Thr Gln Ser Gly Ser Pro Trp Thr Leu Ile Gln His Arg
275 280 285
Lys Asp Gly Ser Gln Asp Phe Asn Glu Thr Trp Glu Asn Tyr Glu Lys
290 295 300
Gly Phe Gly Arg Leu Asp Gly Glu Phe Trp Leu Gly Leu Glu Lys Ile
305 310 315 320
Tyr Ala Ile Val Gln Gln Ser Asn Tyr Ile Leu Arg Leu Glu Leu Gln
325 330 335
Asp Trp Lys Asp Ser Lys His Tyr Val Glu Tyr Ser Phe His Leu Gly
340 345 350
Ser His Glu Thr Asn Tyr Thr Leu His Val Ala Glu Ile Ala Gly Asn
355 360 365
Ile Pro Gly Ala Leu Pro Glu His Thr Asp Leu Met Phe Ser Thr Trp
370 375 380
Asn His Arg Ala Lys Gly Gln Leu Tyr Cys Pro Glu Ser Tyr Ser Gly
385 390 395 400
Gly Trp Trp Trp Asn Asp Ile Cys Gly Glu Asn Asn Leu Asn Gly Lys
405 410 415
Tyr Asn Lys Pro Arg Thr Lys Ser Arg Pro Glu Arg Arg Arg Gly Ile
420 425 430
Tyr Trp Arg Pro Gln Ser Arg Lys Leu Tyr Ala Ile Lys Ser Ser Lys
435 440 445
Met Met Leu Gln Pro Thr Thr
450 455
<210> 485
<211> 460
<212> Белок
<213> Homo sapiens
<400> 485
Met Phe Thr Ile Lys Leu Leu Leu Phe Ile Val Pro Leu Val Ile Ser
1 5 10 15
Ser Arg Ile Asp Gln Asp Asn Ser Ser Phe Asp Ser Leu Ser Pro Glu
20 25 30
Pro Lys Ser Arg Phe Ala Met Leu Asp Asp Val Lys Ile Leu Ala Asn
35 40 45
Gly Leu Leu Gln Leu Gly His Gly Leu Lys Asp Phe Val His Lys Thr
50 55 60
Lys Gly Gln Ile Asn Asp Ile Phe Gln Lys Leu Asn Ile Phe Asp Gln
65 70 75 80
Ser Phe Tyr Asp Leu Ser Leu Gln Thr Ser Glu Ile Lys Glu Glu Glu
85 90 95
Lys Glu Leu Arg Arg Thr Thr Tyr Lys Leu Gln Val Lys Asn Glu Glu
100 105 110
Val Lys Asn Met Ser Leu Glu Leu Asn Ser Lys Leu Glu Ser Leu Leu
115 120 125
Glu Glu Lys Ile Leu Leu Gln Gln Lys Val Lys Tyr Leu Glu Glu Gln
130 135 140
Leu Thr Asn Leu Ile Gln Asn Gln Pro Glu Thr Pro Glu His Pro Glu
145 150 155 160
Val Thr Ser Leu Lys Thr Phe Val Glu Lys Gln Asp Asn Ser Ile Lys
165 170 175
Asp Leu Leu Gln Thr Val Glu Asp Gln Tyr Lys Gln Leu Asn Gln Gln
180 185 190
His Ser Gln Ile Lys Glu Ile Glu Asn Gln Leu Arg Arg Thr Ser Ile
195 200 205
Gln Glu Pro Thr Glu Ile Ser Leu Ser Ser Lys Pro Arg Ala Pro Arg
210 215 220
Thr Thr Pro Phe Leu Gln Leu Asn Glu Ile Arg Asn Val Lys His Asp
225 230 235 240
Gly Ile Pro Ala Glu Cys Thr Thr Ile Tyr Asn Arg Gly Glu His Thr
245 250 255
Ser Gly Met Tyr Ala Ile Arg Pro Ser Asn Ser Gln Val Phe His Val
260 265 270
Tyr Cys Asp Val Ile Ser Gly Ser Pro Trp Thr Leu Ile Gln His Arg
275 280 285
Ile Asp Gly Ser Gln Asn Phe Asn Glu Thr Trp Glu Asn Tyr Lys Tyr
290 295 300
Gly Phe Gly Arg Leu Asp Gly Glu Phe Trp Leu Gly Leu Glu Lys Ile
305 310 315 320
Tyr Ser Ile Val Lys Gln Ser Asn Tyr Val Leu Arg Ile Glu Leu Glu
325 330 335
Asp Trp Lys Asp Asn Lys His Tyr Ile Glu Tyr Ser Phe Tyr Leu Gly
340 345 350
Asn His Glu Thr Asn Tyr Thr Leu His Leu Val Ala Ile Thr Gly Asn
355 360 365
Val Pro Asn Ala Ile Pro Glu Asn Lys Asp Leu Val Phe Ser Thr Trp
370 375 380
Asp His Lys Ala Lys Gly His Phe Asn Cys Pro Glu Gly Tyr Ser Gly
385 390 395 400
Gly Trp Trp Trp His Asp Glu Cys Gly Glu Asn Asn Leu Asn Gly Lys
405 410 415
Tyr Asn Lys Pro Arg Ala Lys Ser Lys Pro Glu Arg Arg Arg Gly Leu
420 425 430
Ser Trp Lys Ser Gln Asn Gly Arg Leu Tyr Ser Ile Lys Ser Thr Lys
435 440 445
Met Leu Ile His Pro Thr Asp Ser Glu Ser Phe Glu
450 455 460
<210> 486
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 486
caggtacagc tgcagcagtc gggcgcagga ctgttgaagc cttcggagac cctgtccctc 60
acctgcactg tctatggtgg atccttcagt attcatcact ggacctggat ccgccatccc 120
ccagggaagg ggctggagtg gattggggag atcaatcatc gtggaagcac caactacaac 180
ccgtccctca agagtcgagt caccatatca atagacacgt ccaagaacca gttctccctg 240
aagctgagcg ctgtgaccgc cgcggacacg gctgtatatt actgtgcgag aggcttacga 300
tttttggact ggttatcgtc ctactttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 487
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 487
Gln Val Gln Leu Gln Gln Ser Gly Ala Gly Leu Leu Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Tyr Gly Gly Ser Phe Ser Ile His
20 25 30
His Trp Thr Trp Ile Arg His Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Asn His Arg Gly Ser Thr Asn Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Ile Asp Thr Ser Lys Asn Gln Phe Ser Leu
65 70 75 80
Lys Leu Ser Ala Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Leu Arg Phe Leu Asp Trp Leu Ser Ser Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 488
<211> 130
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 488
Met His His His His His His Gly Pro Val Gln Ser Lys Ser Pro Arg
1 5 10 15
Phe Ala Ser Trp Asp Glu Met Asn Val Leu Ala His Gly Leu Leu Gln
20 25 30
Leu Gly Gln Gly Leu Arg Glu His Ala Glu Arg Thr Arg Ser Gln Leu
35 40 45
Asn Ala Leu Glu Arg Arg Leu Ser Ala Cys Gly Ser Ala Cys Gln Gly
50 55 60
Thr Glu Gly Ser Thr Ala Leu Pro Leu Ala Pro Glu Ser Arg Val Asp
65 70 75 80
Pro Glu Val Leu His Ser Leu Gln Thr Gln Leu Lys Ala Gln Asn Ser
85 90 95
Arg Ile Gln Gln Leu Phe His Lys Val Ala Gln Gln Gln Arg His Leu
100 105 110
Glu Lys Gln His Leu Arg Ile Gln Arg Leu Gln Ser Gln Val Gly Leu
115 120 125
Leu Asp
130
<210> 489
<211> 444
<212> ДНК
<213> Macaca fascicularis
<400> 489
atgcgcggtg ctccgacggc cggagcagcc ctgatgctct gcgtcgccac ggccgtgctg 60
ctgagagctc agggcggccc ggtgcagtcc aagtctccgc gctttgcgtc ctgggacgag 120
atgaatgtcc tggcgcacgg actcctgcag ctaggccagg ggctgcgcga acacgcggag 180
cgcacccgca gtcagctgaa cgcgctggag cggcgcctca gcgcttgcgg gtctgcctgc 240
cagggaaccg aggggtccac cgccctcccg ttagcccctg agagccgggt ggaccctgag 300
gtccttcaca gcctgcagac acaactcaag gctcagaaca gcaggatcca gcaactcttc 360
cacaaggtgg cccagcagca gcggcacctg gagaagcagc acctgcgaat tcagcgtctg 420
caaagccagg ttggcctcct ggac 444
<210> 490
<211> 148
<212> Белок
<213> Macaca fascicularis
<400> 490
Met Arg Gly Ala Pro Thr Ala Gly Ala Ala Leu Met Leu Cys Val Ala
1 5 10 15
Thr Ala Val Leu Leu Arg Ala Gln Gly Gly Pro Val Gln Ser Lys Ser
20 25 30
Pro Arg Phe Ala Ser Trp Asp Glu Met Asn Val Leu Ala His Gly Leu
35 40 45
Leu Gln Leu Gly Gln Gly Leu Arg Glu His Ala Glu Arg Thr Arg Ser
50 55 60
Gln Leu Asn Ala Leu Glu Arg Arg Leu Ser Ala Cys Gly Ser Ala Cys
65 70 75 80
Gln Gly Thr Glu Gly Ser Thr Ala Leu Pro Leu Ala Pro Glu Ser Arg
85 90 95
Val Asp Pro Glu Val Leu His Ser Leu Gln Thr Gln Leu Lys Ala Gln
100 105 110
Asn Ser Arg Ile Gln Gln Leu Phe His Lys Val Ala Gln Gln Gln Arg
115 120 125
His Leu Glu Lys Gln His Leu Arg Ile Gln Arg Leu Gln Ser Gln Val
130 135 140
Gly Leu Leu Asp
145
<210> 491
<211> 444
<212> ДНК
<213> Macaca mulatta
<400> 491
atgcgcggtg ctccgacggc cggagcagcc ctgatgctct gcgtcgccac ggccgtgctg 60
ctgagagctc agggcggccc ggtgcagtcc aagtctccgc gcttcgcgtc ctgggacgag 120
atgaatgtcc tggcgcacgg actcctgcag ctaggccagg ggctgcgcga acacgcggag 180
cgcacccgca gtcagctgaa cgcgctggag cggcgcctca gcgcttgcgg gtctgcctgc 240
cagggaaccg aggggtccac cgccctcccg ttagcccctg agagccgggt ggaccctgag 300
gtccttcaca gcctgcagac acaactcaag gctcagaaca gcaggatcca gcaactcttc 360
cacaaggtgg cccagcagca gcggcacctg gagaagcagc acctgcgaat tcagcgtctg 420
caaagccagg ttggcctcct ggac 444
<210> 492
<211> 148
<212> Белок
<213> Macaca mulatta
<400> 492
Met Arg Gly Ala Pro Thr Ala Gly Ala Ala Leu Met Leu Cys Val Ala
1 5 10 15
Thr Ala Val Leu Leu Arg Ala Gln Gly Gly Pro Val Gln Ser Lys Ser
20 25 30
Pro Arg Phe Ala Ser Trp Asp Glu Met Asn Val Leu Ala His Gly Leu
35 40 45
Leu Gln Leu Gly Gln Gly Leu Arg Glu His Ala Glu Arg Thr Arg Ser
50 55 60
Gln Leu Asn Ala Leu Glu Arg Arg Leu Ser Ala Cys Gly Ser Ala Cys
65 70 75 80
Gln Gly Thr Glu Gly Ser Thr Ala Leu Pro Leu Ala Pro Glu Ser Arg
85 90 95
Val Asp Pro Glu Val Leu His Ser Leu Gln Thr Gln Leu Lys Ala Gln
100 105 110
Asn Ser Arg Ile Gln Gln Leu Phe His Lys Val Ala Gln Gln Gln Arg
115 120 125
His Leu Glu Lys Gln His Leu Arg Ile Gln Arg Leu Gln Ser Gln Val
130 135 140
Gly Leu Leu Asp
145
Claims (19)
1. Выделенное антитело человека или его антигенсвязывающий фрагмент, которые специфически связываются с ангиопоэтин-подобным белком 4 человека (hANGPTL4), содержащее
три определяющие комплементарность области тяжелой цепи HCDR1/HCDR2/HCDR3 и
три определяющие комплементарность области легкой цепи LCDR1/LCDR2/LCDR3,
где аминокислотные последовательности HCDR1, HCDR2 и HCDR3 содержат SEQ ID NO: 28, 30 и 32, соответственно; и
аминокислотные последовательности LCDR1, LCDR2 и LCDR3 содержат SEQ ID NO: 36, 38 и 40, соответственно.
три определяющие комплементарность области тяжелой цепи HCDR1/HCDR2/HCDR3 и
три определяющие комплементарность области легкой цепи LCDR1/LCDR2/LCDR3,
где аминокислотные последовательности HCDR1, HCDR2 и HCDR3 содержат SEQ ID NO: 28, 30 и 32, соответственно; и
аминокислотные последовательности LCDR1, LCDR2 и LCDR3 содержат SEQ ID NO: 36, 38 и 40, соответственно.
2. Выделенное антитело человека или антигенсвязывающий фрагмент антитела по п. 1, которые специфически связываются с ангиопоэтин-подобным белком 4 человека (hANGPTL4), содержащие вариабельную область тяжелой цепи (HCVR) с аминокислотной последовательностью, выбранной из группы, состоящей из SEQ ID NO: 487, 26, 42 и 46.
3. Антитело или антигенсвязывающий фрагмент по п. 2, дополнительно содержащие вариабельную область легкой цепи (LCVR) с аминокислотной последовательностью, выбранной из группы, состоящей из SEQ ID NO: 44, 34 и 48.
4. Антитело или антигенсвязывающий фрагмент по п. 1, содержащие пару последовательностей HCVR/LCVR с аминокислотной последовательностью SEQ ID NO: 487/44, 26/34, 42/44 и 46/48.
5. Антитело или антигенсвязывающий фрагмент по п. 1, содержащие пару последовательностей HCVR/LCVR с аминокислотной последовательностью SEQ ID NO: 487/44.
6. Выделенное антитело человека или его антигенсвязывающий фрагмент, которые специфически связываются с hANGPTL4, содержащие пару последовательностей HCVR/LCVR с аминокислотной последовательностью, выбранной из группы, состоящей из SEQ ID NO: 487/44, 26/34, 42/44 и 46/48.
7. Антитело или антигенсвязывающий фрагмент по любому из пп. 1-5, где антитело или антигенсвязывающий фрагмент проявляют перекрестную реактивность по отношению к ANGPTL4 яванского макака и ANGPTL4 макака-резуса.
8. Выделенная молекула нуклеиновой кислоты, кодирующая антитело или антигенсвязывающий фрагмент по любому из предшествующих пунктов.
9. Вектор экспрессии, содержащий молекулу нуклеиновой кислоты по п. 8.
10. Способ получения антитела против hANGPTL4 или его антигенсвязывающего фрагмента, включающий
выращивание клетки-хозяина, содержащей вектор экспрессии по п. 9 в условиях, обеспечивающих получение антитела или его фрагмента, и
извлечение антитела или его фрагмента, полученного таким образом.
выращивание клетки-хозяина, содержащей вектор экспрессии по п. 9 в условиях, обеспечивающих получение антитела или его фрагмента, и
извлечение антитела или его фрагмента, полученного таким образом.
11. Фармацевтическая композиция для профилактики или лечения заболевания или расстройства, которое можно предотвратить, облегчить, сократить или ингибировать посредством сокращения или ингибирования активности ANGPTL4, содержащая
антитело или антигенсвязывающий фрагмент по любому из пп. 1-7, и
фармацевтически приемлемый носитель.
антитело или антигенсвязывающий фрагмент по любому из пп. 1-7, и
фармацевтически приемлемый носитель.
12. Фармацевтическая композиция по п. 11, дополнительно содержащая одно или несколько дополнительных терапевтических средств, выбранных из
ингибитора редуктазы HMG-CoA; ингибитора захвата холестерина или повторного всасывания желчной кислоты, или их обоих;
средства, которое усиливает катаболизм липопротеинов;
средства, которое уменьшает количество случаев несмертельного инфаркта миокарда; и
активатора фактора транскрипции LXR.
ингибитора редуктазы HMG-CoA; ингибитора захвата холестерина или повторного всасывания желчной кислоты, или их обоих;
средства, которое усиливает катаболизм липопротеинов;
средства, которое уменьшает количество случаев несмертельного инфаркта миокарда; и
активатора фактора транскрипции LXR.
13. Фармацевтическая композиция по п. 11, дополнительно содержащая одно или несколько дополнительных терапевтических средств, выбранных из статина, ниацина, фибрата и антитела против PCSK9.
14. Способ профилактики или лечения заболевания или нарушения, которое можно предотвратить, облегчить, сократить или ингибировать посредством сокращения или ингибирования активности ANGPTL4, включающий
введение нуждающемуся в этом индивидууму терапевтически эффективного количества фармацевтической композиции по любому из пп. 11-13.
введение нуждающемуся в этом индивидууму терапевтически эффективного количества фармацевтической композиции по любому из пп. 11-13.
15. Способ по п. 14, где заболевание или нарушение выбрано из гипертриглицеридемии, гиперхолестеринемии, хиломикронемии, атерогенной дислипидемии, сердечно-сосудистого заболевания или нарушения, острого панкреатита, неалкогольного стеатогепатита (NASH), диабета и ожирения.
16. Антитело или антигенсвязывающий фрагмент по п. 1 для применения для облегчения, сокращения, ингибирования или профилактики ANGPTL4-опосредованного заболевания или нарушения.
17. Антитело или антигенсвязывающий фрагмент по п. 16, где ANGPTL4-опосредованное заболевание или нарушение выбрано из гипертриглицеридемии, гиперхолестеринемии, хиломикронемии, атерогенной дислипидемии, сердечно-сосудистого заболевания или нарушения, острого панкреатита, неалкогольного стеатогепатита (NASH), диабета и ожирения.
18. Применение антитела или его антигенсвязывающего фрагмента по любому из пп. 1-7 для получения лекарственного средства для облегчения, сокращения, ингибирования или профилактики ANGPTL4-опосредованного заболевания или нарушения.
19. Применение по п. 18, где ANGPTL4-опосредованное заболевание или нарушение выбрано из гипертриглицеридемии, гиперхолестеринемии, хиломикронемии, атерогенной дислипидемии, сердечно-сосудистого заболевания или нарушения, острого панкреатита, неалкогольного стеатогепатита (NASH), диабета и ожирения.
Applications Claiming Priority (11)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US29009209P | 2009-12-24 | 2009-12-24 | |
| US61/290,092 | 2009-12-24 | ||
| US30635910P | 2010-02-19 | 2010-02-19 | |
| US61/306,359 | 2010-02-19 | ||
| US32831610P | 2010-04-27 | 2010-04-27 | |
| US61/328,316 | 2010-04-27 | ||
| US34927310P | 2010-05-28 | 2010-05-28 | |
| US61/349,273 | 2010-05-28 | ||
| US35612610P | 2010-06-18 | 2010-06-18 | |
| US61/356,126 | 2010-06-18 | ||
| PCT/US2010/061987 WO2011079257A2 (en) | 2009-12-24 | 2010-12-23 | Human antibodies to human angiopoietin-like protein 4 |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| RU2012131547A RU2012131547A (ru) | 2014-01-27 |
| RU2580045C2 true RU2580045C2 (ru) | 2016-04-10 |
Family
ID=43629324
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| RU2012131547/10A RU2580045C2 (ru) | 2009-12-24 | 2010-12-23 | Антитела человека против ангиопоэтин-подобного белка 4 человека |
Country Status (29)
| Country | Link |
|---|---|
| US (2) | US8354103B2 (ru) |
| EP (1) | EP2516466B1 (ru) |
| JP (2) | JP6000855B2 (ru) |
| KR (1) | KR101842570B1 (ru) |
| CN (1) | CN102741284A (ru) |
| AR (1) | AR079708A1 (ru) |
| AU (1) | AU2010336369C1 (ru) |
| BR (1) | BR112012017765A2 (ru) |
| CA (1) | CA2785158C (ru) |
| CY (1) | CY1118898T1 (ru) |
| DK (1) | DK2516466T3 (ru) |
| ES (1) | ES2628135T3 (ru) |
| HR (1) | HRP20170524T1 (ru) |
| HU (1) | HUE034722T2 (ru) |
| IL (1) | IL220402A (ru) |
| JO (1) | JO3274B1 (ru) |
| LT (1) | LT2516466T (ru) |
| MX (1) | MX337082B (ru) |
| NZ (1) | NZ600768A (ru) |
| PL (1) | PL2516466T3 (ru) |
| PT (1) | PT2516466T (ru) |
| RS (1) | RS56009B1 (ru) |
| RU (1) | RU2580045C2 (ru) |
| SG (1) | SG181784A1 (ru) |
| SI (1) | SI2516466T1 (ru) |
| TW (1) | TWI488643B (ru) |
| UY (1) | UY33144A (ru) |
| WO (1) | WO2011079257A2 (ru) |
| ZA (1) | ZA201204507B (ru) |
Cited By (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| RU2783685C2 (ru) * | 2019-08-29 | 2022-11-15 | Ремеджен Ко., Лтд. | Анти-pd-l1 антитело и его применение |
Families Citing this family (28)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2012101251A1 (en) | 2011-01-28 | 2012-08-02 | Sanofi | Human antibodies to pcsk9 for use in methods of treatment based on particular dosage regimens |
| AR087305A1 (es) | 2011-07-28 | 2014-03-12 | Regeneron Pharma | Formulaciones estabilizadas que contienen anticuerpos anti-pcsk9, metodo de preparacion y kit |
| JP2014533930A (ja) * | 2011-09-19 | 2014-12-18 | カイマブ・リミテッド | 免疫グロブリン遺伝子多様性の操作およびマルチ抗体治療薬 |
| WO2013155512A2 (en) * | 2012-04-13 | 2013-10-17 | The Johns Hopkins University | Treatment of ischemic retinopathies |
| US9255154B2 (en) | 2012-05-08 | 2016-02-09 | Alderbio Holdings, Llc | Anti-PCSK9 antibodies and use thereof |
| TWI641687B (zh) | 2012-05-29 | 2018-11-21 | 美商再生元醫藥公司 | 生產細胞株增強子 |
| MX363213B (es) | 2012-08-13 | 2019-03-15 | Regeneron Pharma | Anticuerpos anti-pcsk9 con características de unión dependientes del ph. |
| GB201301313D0 (en) * | 2013-01-25 | 2013-03-06 | Univ Singapore | Respiratory Tract Infections |
| WO2014200898A2 (en) * | 2013-06-10 | 2014-12-18 | Merck Sharp & Dohme Corp. | Cmv neutralizing antigen binding proteins |
| KR101538787B1 (ko) * | 2013-06-27 | 2015-07-22 | 인하대학교 산학협력단 | 췌장염 진단용 바이오 마커 조성물 |
| WO2016020882A2 (en) * | 2014-08-07 | 2016-02-11 | Novartis Ag | Angiopoetin-like 4 (angptl4) antibodies and methods of use |
| PT3177642T (pt) * | 2014-08-07 | 2022-02-14 | Novartis Ag | Anticorpos semelhantes a angiopoietina 4 e métodos de uso |
| US9556225B2 (en) * | 2014-10-15 | 2017-01-31 | Uniao Quimica Farmaceutica Nacional S/A | Peptide stimulator of cell survival and proliferation |
| WO2016181409A1 (en) | 2015-05-11 | 2016-11-17 | Cadila Healthcare Limited | Saroglitazar magnesium for the treatment of chylomicronemia syndrome - |
| WO2017011929A1 (zh) * | 2015-07-17 | 2017-01-26 | 北京大学第一医院 | 检测血清血管生成素样蛋白2含量的物质在制备检测肝脏炎症和纤维化程度产品中的应用 |
| TW201713690A (zh) | 2015-08-07 | 2017-04-16 | 再生元醫藥公司 | 抗angptl8抗體及其用途 |
| CN107922507B (zh) | 2015-08-18 | 2022-04-05 | 瑞泽恩制药公司 | 抗pcsk9抑制性抗体用来治疗接受脂蛋白单采的高脂血症患者 |
| WO2017064635A2 (en) | 2015-10-14 | 2017-04-20 | Cadila Healthcare Limited | Pyrrole compound, compositions and process for preparation thereof |
| WO2018094112A1 (en) | 2016-11-17 | 2018-05-24 | Regeneron Pharmaceuticals, Inc. | Methods of treating obesity with anti-angptl8 antibodies |
| US11046764B2 (en) | 2017-01-03 | 2021-06-29 | Trellis Bioscience, Llc | Native human antibodies for immune checkpoint modulation targets TIM-3 and B7-H3 |
| US20190031774A1 (en) | 2017-06-09 | 2019-01-31 | Sanofi Biotechnology | Methods for treating hyperlipidemia in diabetic patients by administering a pcsk9 inhibitor |
| CN114008079A (zh) * | 2019-03-20 | 2022-02-01 | 贾夫林肿瘤学公司 | 抗adam12抗体和嵌合抗原受体以及包含其的组合物和方法 |
| CA3164050A1 (en) * | 2019-12-09 | 2021-06-17 | Empirico Inc. | Oligonucleotides for treatment of angiopoietin like 4 (angptl4) related diseases |
| PL3872091T3 (pl) | 2020-02-26 | 2023-12-27 | Vir Biotechnology, Inc. | Przeciwciała przeciw sars-cov-2 |
| WO2022006555A2 (en) * | 2020-07-02 | 2022-01-06 | Gigagen, Inc. | BISPECIFIC ANTIGEN BINDING PROTEINS TARGETING PD-L1 AND TGF-β AND METHODS OF USE |
| JP2023533253A (ja) * | 2020-07-02 | 2023-08-02 | マブリティクス, インコーポレイテッド | 細胞結合タンパク質および使用の方法 |
| WO2023239307A1 (en) * | 2022-06-10 | 2023-12-14 | Nanyang Technological University | Methods of treating liver fibrosis, inflammation or associated diseases using an angptl4 antagonist |
| TW202415409A (zh) * | 2022-08-02 | 2024-04-16 | 中國商諾納生物(蘇州)有限公司 | Msln抗體藥物複合體 |
Citations (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2007109307A2 (en) * | 2006-03-20 | 2007-09-27 | Curagen Corporation | Antibodies directed to angiopoietin-like protein 4 and uses thereof |
| RU2007127842A (ru) * | 2004-12-21 | 2009-01-27 | Астразенека Аб (Se) | Антитела против ангиопоэтина-2 и их применение |
Family Cites Families (8)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US7070959B1 (en) | 1999-06-08 | 2006-07-04 | Regeneron Pharmaceuticals, Inc. | Modified chimeric polypeptides with improved pharmacokinetic properties |
| US20040101920A1 (en) | 2002-11-01 | 2004-05-27 | Czeslaw Radziejewski | Modification assisted profiling (MAP) methodology |
| US7850962B2 (en) | 2004-04-20 | 2010-12-14 | Genmab A/S | Human monoclonal antibodies against CD20 |
| ZA200705980B (en) * | 2005-01-07 | 2009-01-28 | Lexicon Pharmaceuticals Inc | Monoclonal antibodies against angiopoietin-like protein 4 (ANGPTL4) |
| ES2400666T5 (es) | 2005-12-16 | 2016-03-10 | Regeneron Pharmaceuticals, Inc. | Uso terapéutico de un antagonista de DII4 y un inhibidor de VEGF para inhibir el crecimiento tumoral |
| MY159787A (en) | 2006-06-02 | 2017-01-31 | Regeneron Pharma | High affinity antibodies to human il-6 receptor |
| RU2448979C2 (ru) | 2006-12-14 | 2012-04-27 | Ридженерон Фармасьютикалз, Инк. | Антитела человека к дельта-подобному лиганду-4 человека |
| JO3672B1 (ar) | 2008-12-15 | 2020-08-27 | Regeneron Pharma | أجسام مضادة بشرية عالية التفاعل الكيماوي بالنسبة لإنزيم سبتيليسين كنفرتيز بروبروتين / كيكسين نوع 9 (pcsk9). |
-
2010
- 2010-12-16 JO JOP/2010/0453A patent/JO3274B1/ar active
- 2010-12-22 AR ARP100104893A patent/AR079708A1/es unknown
- 2010-12-22 UY UY0001033144A patent/UY33144A/es unknown
- 2010-12-23 US US12/977,361 patent/US8354103B2/en active Active
- 2010-12-23 CN CN2010800628627A patent/CN102741284A/zh active Pending
- 2010-12-23 WO PCT/US2010/061987 patent/WO2011079257A2/en active Application Filing
- 2010-12-23 EP EP10798240.7A patent/EP2516466B1/en active Active
- 2010-12-23 RS RS20170522A patent/RS56009B1/sr unknown
- 2010-12-23 RU RU2012131547/10A patent/RU2580045C2/ru not_active IP Right Cessation
- 2010-12-23 PT PT107982407T patent/PT2516466T/pt unknown
- 2010-12-23 SG SG2012044871A patent/SG181784A1/en unknown
- 2010-12-23 BR BR112012017765A patent/BR112012017765A2/pt not_active Application Discontinuation
- 2010-12-23 JP JP2012546229A patent/JP6000855B2/ja active Active
- 2010-12-23 KR KR1020127019046A patent/KR101842570B1/ko active IP Right Grant
- 2010-12-23 AU AU2010336369A patent/AU2010336369C1/en active Active
- 2010-12-23 NZ NZ600768A patent/NZ600768A/en not_active IP Right Cessation
- 2010-12-23 TW TW099145406A patent/TWI488643B/zh not_active IP Right Cessation
- 2010-12-23 PL PL10798240T patent/PL2516466T3/pl unknown
- 2010-12-23 DK DK10798240.7T patent/DK2516466T3/en active
- 2010-12-23 SI SI201031459A patent/SI2516466T1/sl unknown
- 2010-12-23 LT LTEP10798240.7T patent/LT2516466T/lt unknown
- 2010-12-23 ES ES10798240.7T patent/ES2628135T3/es active Active
- 2010-12-23 HU HUE10798240A patent/HUE034722T2/en unknown
- 2010-12-23 MX MX2012007460A patent/MX337082B/es active IP Right Grant
- 2010-12-23 CA CA2785158A patent/CA2785158C/en active Active
-
2012
- 2012-06-14 IL IL220402A patent/IL220402A/en not_active IP Right Cessation
- 2012-06-19 ZA ZA2012/04507A patent/ZA201204507B/en unknown
- 2012-12-12 US US13/712,268 patent/US9120851B2/en active Active
-
2015
- 2015-05-11 JP JP2015096446A patent/JP2015145424A/ja active Pending
-
2017
- 2017-03-31 HR HRP20170524TT patent/HRP20170524T1/hr unknown
- 2017-05-17 CY CY20171100521T patent/CY1118898T1/el unknown
Patent Citations (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| RU2007127842A (ru) * | 2004-12-21 | 2009-01-27 | Астразенека Аб (Se) | Антитела против ангиопоэтина-2 и их применение |
| WO2007109307A2 (en) * | 2006-03-20 | 2007-09-27 | Curagen Corporation | Antibodies directed to angiopoietin-like protein 4 and uses thereof |
Cited By (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| RU2783685C2 (ru) * | 2019-08-29 | 2022-11-15 | Ремеджен Ко., Лтд. | Анти-pd-l1 антитело и его применение |
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| RU2580045C2 (ru) | Антитела человека против ангиопоэтин-подобного белка 4 человека | |
| US20220403016A1 (en) | Anti-angptl3 antibodies and uses thereof | |
| JP5318965B2 (ja) | Pcsk9に対する高親和性ヒト抗体 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| MM4A | The patent is invalid due to non-payment of fees |
Effective date: 20201224 |