KR20150133768A - 근육 이영양증의 치료를 위한 개선된 조성물 - Google Patents
근육 이영양증의 치료를 위한 개선된 조성물 Download PDFInfo
- Publication number
- KR20150133768A KR20150133768A KR1020157029546A KR20157029546A KR20150133768A KR 20150133768 A KR20150133768 A KR 20150133768A KR 1020157029546 A KR1020157029546 A KR 1020157029546A KR 20157029546 A KR20157029546 A KR 20157029546A KR 20150133768 A KR20150133768 A KR 20150133768A
- Authority
- KR
- South Korea
- Prior art keywords
- synthetic
- artificial sequence
- dmd
- dna
- rna
- Prior art date
Links
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/113—Non-coding nucleic acids modulating the expression of genes, e.g. antisense oligonucleotides; Antisense DNA or RNA; Triplex- forming oligonucleotides; Catalytic nucleic acids, e.g. ribozymes; Nucleic acids used in co-suppression or gene silencing
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K31/00—Medicinal preparations containing organic active ingredients
- A61K31/70—Carbohydrates; Sugars; Derivatives thereof
- A61K31/7088—Compounds having three or more nucleosides or nucleotides
- A61K31/7125—Nucleic acids or oligonucleotides having modified internucleoside linkage, i.e. other than 3'-5' phosphodiesters
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K9/00—Medicinal preparations characterised by special physical form
- A61K9/0012—Galenical forms characterised by the site of application
- A61K9/0019—Injectable compositions; Intramuscular, intravenous, arterial, subcutaneous administration; Compositions to be administered through the skin in an invasive manner
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P1/00—Drugs for disorders of the alimentary tract or the digestive system
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P21/00—Drugs for disorders of the muscular or neuromuscular system
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P21/00—Drugs for disorders of the muscular or neuromuscular system
- A61P21/02—Muscle relaxants, e.g. for tetanus or cramps
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/30—Chemical structure
- C12N2310/33—Chemical structure of the base
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2320/00—Applications; Uses
- C12N2320/30—Special therapeutic applications
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Engineering & Computer Science (AREA)
- Genetics & Genomics (AREA)
- General Health & Medical Sciences (AREA)
- Organic Chemistry (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Molecular Biology (AREA)
- Biomedical Technology (AREA)
- Medicinal Chemistry (AREA)
- Public Health (AREA)
- Pharmacology & Pharmacy (AREA)
- Veterinary Medicine (AREA)
- Animal Behavior & Ethology (AREA)
- Biochemistry (AREA)
- General Engineering & Computer Science (AREA)
- Zoology (AREA)
- Wood Science & Technology (AREA)
- Biotechnology (AREA)
- Epidemiology (AREA)
- General Chemical & Material Sciences (AREA)
- Nuclear Medicine, Radiotherapy & Molecular Imaging (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Physics & Mathematics (AREA)
- Plant Pathology (AREA)
- Microbiology (AREA)
- Biophysics (AREA)
- Neurology (AREA)
- Orthopedic Medicine & Surgery (AREA)
- Physical Education & Sports Medicine (AREA)
- Pain & Pain Management (AREA)
- Dermatology (AREA)
- Pharmaceuticals Containing Other Organic And Inorganic Compounds (AREA)
- Medicines That Contain Protein Lipid Enzymes And Other Medicines (AREA)
- Peptides Or Proteins (AREA)
- Medicines Containing Material From Animals Or Micro-Organisms (AREA)
Abstract
인간 디스트로핀 유전자 내의 선택된 표적 부위에 결합하여 엑손 스키핑을 유도할 수 있는 안티센스 분자, 예컨대 에테플러센을 투여함으로써 근육 이영양증을 치료하는 개선된 조성물 및 방법이 기재된다.
Description
관련 출원
본 특허 출원은 2013년 3월 15일에 제출된 미국 가특허 출원 일련 번호 61/793,463의 이익을 주장한다. 상기 언급한 가특허 출원의 전문이 본원에 참조로 포함된다.
발명의 분야
본 발명은 환자에서 근육 이영양증의 치료를 위한 개선된 방법에 관한 것이다. 본 발명은 또한 인간 디스트로핀 유전자에서 엑손 스키핑을 용이하게 하는 데 적합한 조성물을 제공한다.
안티센스 기술은 다양한 상이한 수준 (전사, 스플라이싱, 안정화, 번역)에서 유전자 발현에 영향을 주는 다양한 화학을 사용하여 개발되고 있다. 많은 그 연구는 폭넓은 범위의 적응증에서 비정상적 또는 질환-연관된 유전자를 수정 또는 보정하기 위한 안티센스 화합물의 사용에 초점을 맞추었다. 안티센스 분자는 특이성을 갖는 유전자 발현을 억제할 수 있고, 이 때문에 유전자 발현의 조절제로서의 올리고뉴클레오티드에 관심을 갖는 많은 연구는 표적화된 유전자의 발현 또는 시스-작용 요소의 기능을 억제하는 데 초점을 맞추었다. 안티센스 올리고뉴클레오티드는 전형적으로 센스 가닥 (예를 들어, mRNA), 또는 일부 바이러스 RNA 표적의 경우 마이너스-가닥인 RNA에 대해 지정된다. 특이적 유전자 하류-조절의 바람직한 효과를 달성하기 위해, 올리고뉴클레오티드는 일반적으로 표적화된 mRNA의 붕괴를 촉진하거나, mRNA의 번역을 차단하거나, 시스-작용 RNA 요소의 기능을 차단함으로써, 표적 단백질의 신생 합성 또는 바이러스 RNA의 복제를 효과적으로 방지한다.
그러나, 이러한 기술은 넌센스 또는 프레임-이동 돌연변이와 같이 목적이 천연 단백질의 생산을 상향-조절하거나, 번역의 미성숙 종결을 유도하는 돌연변이를 보정하기 위한 것일 경우에는 유용하지 않다. 이들 경우에, 결함성 유전자 전사체는 표적화된 분해 또는 입체적 억제를 겪지 않아야 하며, 따라서 안티센스 올리고뉴클레오티드 화학은 표적 mRNA 붕괴를 촉진하거나 번역을 차단하지 않아야 한다.
다양한 유전적 질환에서, 유전자의 궁극적인 발현에 대한 돌연변이의 효과는 스플라이싱 프로세스 동안 표적화된 엑손 스키핑의 프로세스를 통해 조절될 수 있다. 스플라이싱 프로세스는 프리-mRNA(프리-mRNA)에서의 인접한 엑손-인트론 접합이 매우 근접하게 하고, 인트론의 말단에서 포스포디에스테르 결합의 절단과, 함께 스플라이싱되는 엑손 사이에서 그들의 후속 변형을 수행하는 복잡한 다중-성분 기구에 의해 감독된다. 이 복잡하고 매우 정확한 프로세스는 이어서 스플라이싱 반응에 관련되는 다양한 핵 스플라이싱 인자가 결합하는, 상대적으로 짧고, 반-보존된 RNA 절편인 프리-mRNA에서의 서열 모티프에 의해 매개된다. 스플라이싱 기구가 프리-mRNA 프로세싱에 관련된 모티프를 판독하거나 인식하는 방식을 변화시킴으로써, 차등적으로 스플라이싱된 mRNA 분자를 생성할 수 있다. 그 메커니즘은 확인되지 않았지만, 이제는 대부분의 인간 유전자가 정상적인 유전자 발현 동안 선택적으로 스플라이싱된다는 것이 인지되었다. 베네트 등(Bennett et al.) (미국 특허 제6,210,892호)은 표적 RNA의 RNAse H-매개된 절단을 유도하지 않는 안티센스 올리고뉴클레오티드 유사체를 사용한 야생형 세포 mRNA 프로세싱의 안티센스 조절을 기재하고 있다. 이는 막 스패닝 도메인을 코딩하는 엑손이 결핍된 가용성 TNF 슈퍼패밀리 수용체의 생성을 위한 특이적 엑손이 결핍된 선택적으로 스플라이싱된 mRNA를 생성할 수 있는 유용성을 발견하고 있다 (예를 들어, 문헌 [Sazani, Kole, et al. 2007]에 의해 기재된 바와 같음).
정상적 기능성 단백질이 그 안의 돌연변이로 인해 미성숙하게 종결되는 경우, 안티센스 기술을 통해 일부 기능성 단백질 생성을 복원하는 수단은 스플라이싱 프로세스 동안 개입을 통해 가능한 것으로 나타났으며, 질환-유발 돌연변이와 연관된 엑손이 일부 유전자로부터 특이적으로 결실될 수 있을 경우, 천연 단백질의 유사한 생물학적 특성을 갖거나, 엑손과 된 돌연변이에 의해 유발되는 질환을 개선하는 충분한 생물학적 활성을 갖는 짧아진 단백질 생성물이 때때로 생성될 수 있다 (예를 들어, 문헌 [Sierakowska, Sambade et al. 1996]; [Wilton, Lloyd et al. 1999]; [van Deutekom, Bremmer-Bout et al. 2001]; [Lu, Mann et al. 2003]; [Aartsma-Rus, Janson et al. 2004] 참조). 콜 등(Kole et al.) (미국 특허 제5,627,274호; 제5,916,808호; 제5,976,879호; 및 제5,665,593호)은 표적화된 프리-mRNA의 붕괴를 촉진시키지 않는 변형된 안티센스 올리고뉴클레오티드 유사체를 사용한 비정상적 스플라이싱의 방지 방법을 개시하고 있다. 베네트 등 (미국 특허 제6,210,892호)은 표적 RNA의 RNAse H-매개된 절단을 유도하지 않는 안티센스 올리고뉴클레오티드 유사체를 또한 사용한 야생형 세포 mRNA 프로세싱의 안티센스 조절을 기재하고 있다.
표적화된 엑손 스키핑의 프로세스는 많은 엑손 및 인트론이 있는 긴 유전자에서 특히 유용할 가능성이 있으며, 여기서 엑손의 유전적 구성에 중복이 있거나, 단백질이 하나 이상의 특정 엑손 없이 기능할 수 있다. 다양한 유전자에서의 돌연변이에 의해 유발되는 말단절단과 연관된 유전적 질환의 치료를 위한 유전자 프로세싱을 재지정하기 위한 노력은 (1) 스플라이싱 프로세스에 관련된 요소와 완전히 또는 부분적으로 중복되거나; 또는 (2) 그 요소에서 일어나는 특정 스플라이싱 반응을 정상적으로 매개할 스플라이싱 인자의 결합 및 기능을 방해하는 요소에 충분히 가까운 위치에서 프리-mRNA에 결합하는 안티센스 올리고뉴클레오티드의 사용에 초점을 맞추었다.
뒤시엔느(Duchenne) 근육 이영양증 (DMD)은 단백질 디스트로핀의 발현의 결함에 의해 유발된다. 단백질을 코딩하는 유전자는 2백만개 초과의 뉴클레오티드의 DNA에 걸쳐 퍼져 있는 79개의 엑손을 함유한다. 엑손의 리딩 프레임을 변화시키거나, 정지 코돈을 도입하거나, 프레임 엑손 또는 엑손들로부터의 전체의 제거 또는 하나 이상의 엑손의 복제를 특징으로 하는 임의의 엑손 돌연변이는 기능성 디스트로핀의 생성을 방해하여 DMD를 초래할 잠재성을 갖는다.
질환 개시는 출생시에 증가된 크레아틴 키나제 수준으로서 문서 기록될 수 있으며, 생후 1년 안에 상당한 운동 결손이 존재할 수 있다. 7세 또는 8세까지, 대부분의 DMD 환자는 부자연스러운 걸음걸이가 점차 증가되며, 바닥으로부터 일어서고 계단을 오르는 능력을 상실하며; 10세 내지 14세까지, 대부분 휠체어에 의존하게 된다. DMD는 한결같이 치명적이며; 병에 걸린 개개인은 전형적으로 10대 후반 또는 20대 초반에 호흡 부전 및/또는 심부전으로 사망한다. DMD의 지속적인 진행은 질환의 모든 단계에서 치료적 개입을 허용한다; 그러나, 치료는 현재 글루코코르티코이드로 제한되어 있는데, 이는 체중 증가, 행동 변화, 사춘기 변화, 골다공증, 쿠싱 유사 외견(Cushingoid facies), 성장 억제, 및 백내장을 비롯한 수많은 부작용과 연관되어 있다. 따라서, 이 질환의 기저 원인을 치료하는 보다 나은 치료법을 개발하는 것이 반드시 필요하다.
근육 이영양증의 덜 심각한 형태인 베커(Becker) 근육 이영양증 (BMD)은 돌연변이, 전형적으로 하나 이상의 엑손의 결실이 전체 디스트로핀 전사체를 따라 올바른 리딩 프레임을 초래하여, mRNA의 단백질로의 번역이 미성숙하게 종결될 경우 일어나는 것으로 밝혀졌다. 돌연변이된 디스트로핀 프리-mRNA의 프로세싱에서 상류 및 하류 엑손의 결합이 유전자의 올바른 리딩 프레임을 유지하는 경우, 결과는 베커 표현형을 초래하는, 일부 활성을 보유하는 짧은 내부 결실을 갖는 단백질에 대한 mRNA 코딩이다.
수년 동안, 디스트로핀 단백질의 리딩 프레임을 변경하지 않는 엑손 또는 엑손들의 결실은 BMD 표현형을 일으키는 반면, 프레임-이동을 유발하는 엑손 결실은 DMD를 일으키는 것으로 알려졌다 (Monaco, Bertelson et al. 1988). 일반적으로, 리딩 프레임을 변화시키고, 따라서 적절한 단백질 번역을 방해하는 점 돌연변이 및 엑손 결실을 포함하는 디스트로핀 돌연변이는 DMD를 초래한다. 또한, 일부 BMD 및 DMD 환자는 다중 엑손을 커버하는 엑손 결실을 가짐이 주목되어야 한다.
안티센스 올리고리보뉴클레오티드를 사용한 돌연변이체 디스트로핀 프리-mRNA 스플라이싱의 조절은 시험관내 및 생체내 둘 다에서 보고되었다 (예를 들어, 문헌 [Matsuo, Masumura et al. 1991]; [Takeshima, Nishio et al. 1995]; [Pramono, Takeshima et al. 1996]; [Dunckley, Eperon et al. 1997]; [Dunckley, Manoharan et al. 1998]; [Errington, Mann et al. 2003] 참조).
mdx 마우스 모델에서 특이적 및 재생적 엑손 스키핑의 첫번째 예는 윌톤 등(Wilton et al.)에 의해 보고되었다 (Wilton, Lloyd et al. 1999). 공여자 스플라이스 부위에 안티센스 분자를 지정함으로써, 배양된 세포의 처리 6시간 내에 일정하고 효과적인 엑손 23 스키핑이 디스트로핀 mRNA에서 유도되었다. 윌톤 등은 또한 보다 긴 안티센스 올리고뉴클레오티드를 갖는 마우스 디스트로핀 프리-mRNA의 수용자 영역의 표적화를 기재하고 있다. 인트론 23 공여자 스플라이스 부위에서 지정된 제1 안티센스 올리고뉴클레오티드는 1차 배양된 근모세포에서 일정한 엑손 스키핑을 유도한 반면, 이 화합물은 보다 높은 수준의 디스트로핀을 발현하는 불멸화 세포 배양물에서 훨씬 덜 효과적인 것으로 밝혀졌다. 그러나, 엄밀한 표적화 및 안티센스 올리고뉴클레오티드 설계로, 특이적 엑손 제거의 효율이 거의 한 자릿수로 증가되었다 (Mann, Honeyman et al. 2002).
최근의 연구는 디스트로핀의 부재에 의해 영향을 받는 조직에서 최소의 역효과를 수반하는 지속성 디스트로핀 발현을 달성하는 도전을 다루기 시작했다. DMD를 갖는 4명의 환자에서 엑손 51 (PRO051)로 표적화된 안티센스 올리고뉴클레오티드의 전경골근 내로의 근육내 주입은 임의의 임상적으로 명백한 역효과 없이 엑손 51의 특이적 스키핑을 초래하였다 (Mann, Honeyman et al. 2002; van Deutekom, Janson et al. 2007). mdx 마우스에서 엑손 23으로 표적화된 세포-관통 펩티드에 접합된 안티센스 포스포로디아미데이트 모르폴리노 올리고머 (PPMO)의 전신적 전달을 살펴본 연구는 검출가능한 독성 없이 골격근 및 심근에서 높고 지속된 디스트로핀 단백질 생성을 산출하였다 (Jearawiriyapaisarn, Moulton et al. 2008; Wu, Moulton et al. 2008; Yin, Moulton et al. 2008).
DMD의 치료를 위한 스플라이스 스위칭 올리고뉴클레오티드 (SSO)의 안전성 및 효율을 시험하는 최근의 임상적 시도는 스플라이세오솜의 입체적 봉쇄에 의해 프리-mRNA의 선택적 스플라이싱을 유도하는 SSO 기술에 기초한다 (Cirak et al., 2011; Goemans et al., 2011; Kinali et al., 2009; van Deutekom et al., 2007). 그러나, 이들 성공에도 불구하고, DMD의 치료에 이용가능한 약리학적 선택사항은 제한되어 있다. 주목할 만한 것으로서, 음으로 하전된 포스포로티오에이트 골격을 이용하는 안티센스 올리고뉴클레오티드 (드리사페르센)는 임상 시험에서 단백뇨, 증가된 비뇨기 α1-마이크로글로불린, 혈소판감소증 및 주사 부위 반응, 예컨대 홍반 및 염증과 연관성이 있어 왔다.
본원의 양수인에 의해 개발되고 있는 에테플러센은 시험 그의 안전성 및 효능을 시험하는 임상 연구의 대상이었으며, 임상 개발이 진행중이다. 에테플러센은 드리사페르센과는 구조적으로 뚜렷이 구별되는 올리고뉴클레오티드이다. 구체적으로, 에테플러센의 화학적 골격은 포스포로디아미데이트 모폴리노 (PMO)인 반면, 드리사페르센의 화학적 골격은 2'O-메틸 포스포로티오에이트 (2'-OMe)이다. 이러한 구조적 차이 및 임상 결과에 대한 이들의 잠재적 영향이 최근 기재되었다. 문헌 [Molecular Therapy Nucleic Acids (2014) 3, e152; doi:10.1038/mtna.2014.6 (2014년 3월 11일 온라인 공개) 참조.
에테플러센의 서열은 이전에 기재된 바 있다. 예를 들어, 배타적으로 출원인에 허가된 미국 특허 제7,807,816호 참조. 그러나, 미국 특허 제7,807,816호는 에테플러센에 대한 최적의 투여 스케쥴 및 투여 경로를 명시적으로 논의하지 않는다.
따라서, 환자에서 DMD 및 BMD와 같은 근육 이영양증을 치료하기 위한 개선된 조성물 및 방법에 대한 필요가 남아있다.
발명의 개요
본 발명은, 적어도 부분적으로, 엑손 스키핑 안티센스 올리고뉴클레오티드인 에테플러센의 치료 효과의 강력한 증거에 기초한 것으로, 이는 질환의 기저 원인을 다룸으로써 DMD의 치료에서 중대한 진보를 나타낸다. 엑손 51 스키핑 안티센스 올리고뉴클레오티드인 에테플러센을 이용한 치료가 신규 디스트로핀의 신뢰성 있는 증가 및 6분 보행 시험 (6MWT)으로 측정된 바 안정화된 보행 능력 (예를 들어, 보행의 안정화)을 가져왔다는 신규 발견은 질환의 기간을 변경시킬 잠재성을 강조한다. 유의미하게, 1년에 걸쳐 투여된 576회의 주입에서 약물-관련 유해 사례는 관찰되지 않았다. 다른 엑손에 적용시, 엑손 스키핑 안티센스 올리고뉴클레오티드의 사용은 디스트로핀 유전자 내의 결실로 인해 DMD를 갖는 환자의 대략 70% 내지 80%를 치료할 수 있었다.
따라서, 한 측면에서 본 발명은 뒤시엔느 근육 이영양증의 치료를 필요로 하는 환자에게 약 30 mg/kg의 용량의 에테플러센을 주 1회 투여하는 것을 포함하는, 상기 환자에서 뒤시엔느 근육 이영양증을 치료하는 방법에 관한 것이다. 일부 실시양태에서, 에테플러센은 단일 용량으로 투여된다. 일부 실시양태에서 에테플러센은 정맥내로 투여된다. 일부 실시양태에서, 환자는 디스트로핀 유전자의 엑손 51의 스키핑에 의해 교정될 수 있는 아웃-오브-프레임(out-of-frame) 결실(들)을 갖는다. 일부 실시양태에서, 환자는 소아 환자이다.
또 다른 측면에서, 본 발명은 뒤시엔느 근육 이영양증의 치료를 필요로 하는 환자에게 약 30 mg/kg의 용량의 에테플러센을 주 1회 투여하는 것을 포함하며, 여기서 환자는 제1 용량의 에테플러센 투여 전에 적어도 24주 동안 경구 코르티코스테로이드를 투여받는 것인, 상기 환자에서 뒤시엔느 근육 이영양증을 치료하는 방법에 관한 것이다.
또 다른 측면에서, 본 발명은 뒤시엔느 근육 이영양증의 치료를 필요로 하는 환자에게 단일 용량의 약 30 mg/kg의 에테플러센을 주 1회 정맥내로 투여하는 것을 포함하며, 여기서 상기 환자는 디스트로핀 유전자의 엑손 51의 스키핑에 의해 교정될 수 있는 아웃-오브-프레임 결실(들)을 갖는 환자인, 상기 환자에서 뒤시엔느 근육 이영양증을 치료하는 방법을 제공한다.
본 발명의 다른 실시양태는 뒤시엔느 근육 이영양증의 치료를 필요로 하는 환자에게 약 50 mg/kg의 용량의 에테플러센을 주 1회 투여하는 것을 포함하는, 상기 환자에서 뒤시엔느 근육 이영양증을 치료하는 방법에 관한 것이다. 일부 실시양태에서, 에테플러센은 단일 용량을 투여된다. 일부 실시양태에서, 에테플러센은 정맥내로 투여된다. 일부 실시양태에서 환자는 디스트로핀 유전자의 엑손 51의 스키핑에 의해 교정될 수 있는 아웃-오브-프레임 결실(들)을 갖는다. 일부 실시양태에서, 환자는 소아 환자이다. 또 다른 실시양태에서, 환자는 제1 용량의 에테플러센 투여 전에 적어도 24주 동안 경구 코르티코스테로이드를 투여받는다.
또 다른 측면에서, 본 발명은 뒤시엔느 근육 이영양증의 치료를 필요로 하는 환자에게 단일 용량의 약 50 mg/kg의 에테플러센을 주 1회 정맥내로 투여하는 것을 포함하며, 여기서 상기 환자는 디스트로핀 유전자의 엑손 51의 스키핑에 의해 교정될 수 있는 아웃-오브-프레임 결실(들)을 갖는 환자인, 상기 환자에서 뒤시엔느 근육 이영양증을 치료하는 방법에 관한 것이다.
또 다른 측면에서, 본 발명은 뒤시엔느 근육 이영양증을 갖는 환자에게 약 30 mg/kg의 용량의 에테플러센을 주 1회 투여하는 것을 포함하는, 상기 환자에서 디스트로핀 제조를 증가시키는 방법을 제공한다. 일부 실시양태에서는, 약 50 mg/kg의 용량의 에테플러센을 주 1회 투여한다. 일부 실시양태에서, 에테플러센은 단일 용량으로 투여된다. 일부 실시양태에서, 에테플러센은 정맥내로 투여된다. 다른 실시양태에서, 환자는 디스트로핀 유전자의 엑손 51의 스키핑에 의해 교정될 수 있는 아웃-오브-프레임 결실(들)을 갖는다. 일부 실시양태에서 환자는 소아 환자이다. 다른 실시양태에서, 환자는 제1 용량의 에테플러센 투여 전에 적어도 24주 동안 경구 코르티코스테로이드를 투여받는다.
따라서, 본 발명은, 표적 영역에 특이적으로 혼성화하여 엑손 스키핑을 유도하고 그에 의해 질환을 치료하는, 인간 디스트로핀 유전자의 엑손 내 표적 영역에 상보적인 적어도 10개의 연속적 뉴클레오티드를 포함하는 20 내지 50개 뉴클레오티드 길이의 안티센스 올리고뉴클레오티드를 포함하는 유효량의 조성물을 투여함에 의해 환자에서 뒤시엔느 근육 이영양증 (DMD) 또는 베커 근육 이영양증 (BMD)을 치료하는 방법에 관한 것이다. 한 실시양태에서, 유효량은 대상체에서 디스트로핀-양성 섬유의 수를 정상의 적어도 20%까지 증가시키고, 건강한 동년배와 비교하여 환자에서, 예를 들어 6 MWT에서, 20% 결손으로부터의 보행 거리를 안정화, 유지 또는 개선시키기에 충분한 일정 기간동안 적어도 20 mg/kg이다. 또 다른 실시양태에서, 유효량은 적어도 20 mg/kg 내지 약 30 mg/kg, 약 25 mg/kg 내지 약 30 mg/kg, 또는 약 30 mg/kg 내지 약 50 mg/kg이다. 또 다른 실시양태에서, 유효량은 약 30 mg/kg 또는 약 50 mg/kg이다.
또 다른 측면에서, 대상체에서 디스트로핀-양성 섬유의 수를 적어도 정상의 20%, 약 30%, 약 40%, 약 50%, 약 60%, 약 70%, 약 80%, 약 90%, 약 95%까지 증가시키고, 건강한 동년배와 비교하여 환자에서, 예를 들어 6 MWT에서, 20% 결손으로부터의 보행 거리를 안정화 또는 개선시킬 수 있는 유효량은 적어도 24주, 적어도 36주, 또는 적어도 48주 동안 적어도 20 mg/kg, 약 25 mg/kg, 약 30 mg/kg, 또는 약 30 mg/kg 내지 약 50 mg/kg이다. 한 실시양태에서, 치료는 환자에서 디스트로핀-양성 섬유의 수를 정상의 20-60%, 또는 30-50%까지 증가시킨다. 일부 실시양태에서, 치료는 전신 투여에 의해, 예컨대 매주 1회 주입에 의해 이루어진다. 다른 실시양태에서, 치료는 대상체에 또 다른 치료제, 예컨대 스테로이드를 투여하는 것을 포함한다.
또 다른 측면에서, 본 발명은, 인간 디스트로핀 유전자의 엑손 내 표적 영역에 상보적인 적어도 10개의 연속적 뉴클레오티드를 포함하는 20 내지 50개 뉴클레오티드 길이의 안티센스 올리고뉴클레오티드를 포함하는 약 30 mg/kg 내지 약 50 mg/kg의 조성물을 투여함에 의해 환자에서 DMD 또는 BMD를 치료하는 방법을 제공하며, 여기서 안티센스 올리고뉴클레오티드는 표적 영역에 특이적으로 혼성화하여 엑손 스키핑을 유도하고 그에 의해 대상체를 치료한다. 한 실시양태에서, 안티센스 올리고뉴클레오티드는 실질적으로 비하전된다. 또 다른 실시양태에서, 안티센스 올리고뉴클레오티드는 하나의 서브유닛의 모르폴리노 질소를 인접한 서브유닛의 5' 고리외 탄소에 결합시키는 인-함유 서브유닛간 연결에 의해 연결된 모르폴리노 서브유닛을 포함한다. 또 다른 실시양태에서, 안티센스 올리고뉴클레오티드는 하나의 서브유닛의 모르폴리노 질소를 인접한 서브유닛의 5' 고리외 탄소에 결합시키는 실질적으로 비하전된 인-함유 서브유닛간 연결에 의해 연결된 모르폴리노 서브유닛을 포함한다. 다른 측면에서, 안티센스 올리고뉴클레오티드는 모르폴리노 서브유닛 및 포스포로디아미데이트 서브유닛간 연결을 포함한다.
일부 실시양태에서, 안티센스 올리고뉴클레오티드는 엑손 51, 엑손 50, 엑손 53, 엑손 45, 엑손 46, 엑손 44, 엑손 52, 엑손 55 및 엑손 8로 이루어진 군으로부터 선택되는 인간 디스트로핀 유전자의 엑손 내 표적 영역에 상보적인 적어도 10, 12, 15, 17, 또는 20개의 연속적 뉴클레오티드를 포함하는 길이 20 내지 50개, 30 내지 50개, 또는 20 내지 30개의 뉴클레오티드이다. 한 실시양태에서, 안티센스는 길이 20 내지 50개, 30 내지 50개, 또는 20 내지 30개의 뉴클레오티드이며, 적어도 20 연속적 뉴클레오티드 에테플러센 (서열 1000)을 포함한다. 또 다른 실시양태에서, 안티센스 올리고뉴클레오티드는 길이 20 내지 50개, 30 내지 50개, 또는 20 내지 30개의 뉴클레오티드이며, 서열 1001, 서열 1002, 서열 1003, 서열 1004, 서열 1005, 서열 1006, 서열 1007, 또는 서열 1008로 개시된 안티센스 올리고뉴클레오티드의 적어도 10, 12, 15, 17, 또는 20개의 연속적 뉴클레오티드를 포함한다. 또 다른 실시양태에서, 안티센스 올리고뉴클레오티드는 길이 20 내지 50개, 30 내지 50개, 또는 20 내지 30개의 뉴클레오티드이며, 표 3 및 4에 개시된 뉴클레오티드 서열의 적어도 10, 12, 15, 17, 또는 20개의 연속적 뉴클레오티드를 포함하고, 여기서 안티센스 올리고뉴클레오티드 내의 우라실 염기는 임의로 티민 염기이다.
한 실시양태에서, 조성물은 에테플러센 (서열 1000) 및, 임의로, 제약상 허용되는 담체를 포함한다. 또 다른 실시양태에서, 조성물은 서열 1000-1008, 예컨대 서열 1000, 서열 1001, 서열 1002, 서열 1003, 서열 1004, 서열 1005, 서열 1006, 서열 1007, 및 서열 1008로 이루어진 군으로부터 선택되는 안티센스 올리고뉴클레오티드를 포함한다. 또 다른 실시양태에서, 안티센스 올리고뉴클레오티드는 표 3 및 4에 개시된 뉴클레오티드 서열 중 임의의 하나 또는 조합이며, 여기서 안티센스 올리고뉴클레오티드 내의 우라실 염기는 임의로 티민 염기이다. 일부 측면에서, 안티센스 올리고뉴클레오티드는 안티센스 올리고뉴클레오티드의 활성, 세포 분포 또는 세포 흡수를 증진시키는 하나 이상의 모이어티 또는 접합체, 예컨대 아르기닌-풍부한 펩티드에 화학적으로 연결된다.
또 다른 측면에서, 본 발명은 대상체에서 디스트로핀-양성 섬유의 수를 정상의 적어도 약 20%까지 증가시키고, 건강한 동년배와 비교하여 환자에서, 예를 들어 6 MWT에서, 20% 결손으로부터의 보행 거리를 안정화 또는 개선시키기에 충분한 일정 기간동안 적어도 20 mg/kg의 에테플러센 (서열 1000)을 포함하는 조성물을 투여함으로써 환자에서 DMD 또는 BMD를 치료하는 방법을 제공한다. 또 다른 실시양태에서, 유효량은 적어도 20 mg/kg 내지 약 30 mg/kg, 약 25 mg/kg 내지 약 30 mg/kg, 또는 약 30 mg/kg 내지 약 50 mg/kg의 에테플러센 (서열 1000)을 포함하는 조성물 및, 임의로, 제약상 허용되는 담체, 예컨대 포스페이트-완충 염수이다.
또 다른 측면에서, 대상체에서 디스트로핀-양성 섬유의 수를 정상의 적어도 약 20%, 약 30%, 약 40%, 약 50%, 약 60%, 약 70%, 약 80%, 약 90%, 약 95%까지 증가시키고, 건강한 동년배와 비교하여 환자에서, 예를 들어 6 MWT에서, 20% 결손으로부터의 보행 거리를 안정화 또는 개선시킬 수 있는, 에테플러센 (서열 1000)을 포함하는 조성물의 유효량은 적어도 24주, 적어도 36주, 또는 적어도 48주 동안 적어도 20 mg/kg, 약 25 mg/kg, 약 30 mg/kg, 또는 약 30 mg/kg 내지 약 50 mg/kg이다. 일부 실시양태에서, 본 발명의 안티센스 올리고뉴클레오티드를 이용한 치료는 치료 부재시 예상되는 보행 손실을 둔화시키거나 감소시킨다. 일부 실시양태에서, 본 발명의 안티센스 올리고뉴클레오티드를 이용한 치료는 환자에서 안정한 보행 거리를 안정화, 유지, 또는 증가시킨다. 예를 들어, 치료는 기준선으로부터 3, 5, 6, 7, 8, 9, 10, 15, 20, 25, 30 또는 50 미터 (그 사이의 모든 정수를 포함함)를 초과하도록 환자에서 안정한 보행 거리를 증가시킬 수 있다.
본 발명의 다른 측면은 DMD 환자에서 치료 부재시에 예상되었던 진행성 호흡근 기능장애 및/또는 부전을 둔화시키거나 또는 감소시키는 본 발명의 안티센스 올리고뉴클레오티드, 예컨대 에테플러센을 이용한 치료에 관한 것이다. 한 실시양태에서, 본 발명의 안티센스 올리고뉴클레오티드를 이용한 치료는 치료 부재시에 예상되었던 호흡 보조의 필요성을 감소 또는 제거시킬 수 있다. 한 실시양태에서, 질환의 추세를 추적하는 호흡 기능의 측정, 뿐만 아니라 잠재적 치료적 개입에 대한 평가에는 최대 흡기압 (MIP), 최대 호기압 (MEP) 및 강제 폐활량 (FVC)이 포함된다.
도 1a는 포스포로디아미데이트 연결을 갖는 예시적인 모르폴리노 올리고머 구조를 나타낸다.
도 1b는 본 발명의 실시양태에 따른 아르기닌-풍부 펩티드 및 안티센스 올리고머의 접합체를 나타낸다.
도 1c는 골격 연결이 하나 이상의 양으로 하전된 기를 함유하는 도 1b에서와 같은 접합체를 나타낸다.
도 1d 내지 1g는 d 내지 g로 지정된 예시적인 모르폴리노 올리고뉴클레오티드의 반복 서브유닛 절편을 나타낸다.
도 2는 DMD 환자를 치료하는 연구 설계의 개략적 표현이다. 12명의 DMD 환자를 이중-맹검, 위약-대조 연구, 201에서 3개의 코호트 중 하나에 무작위 배정하였다: 코호트 1, 에테플러센 30 mg/kg/wk; 코호트 2, 에테플러센 50 mg/kg/wk; 및 코호트 3, 위약/지연 에테플러센. 제25주에, 코호트 3 내의 위약-치료 환자를 30 또는 50 mg/kg/주 에테플러센을 이용한 개방-표지 치료로 전환하였다. 개방-표지 연장 연구, 202 하에서 환자들을 그들의 동일한 용량의 에테플러센으로 유지시켰다. 근육 생검. 환자는 기준선에서 이두근 생검을 거쳤으며, 디스트로핀의 분석을 위해 제48주에 삼각근 생검을 거쳤다. 제12주에 (코호트 2의 환자 및 코호트 3의 2명의 환자로부터) 또는 제24주에 (코호트 1의 환자 및 코호트 3의 2명의 환자로부터) 추가의 이두근 생검을 수득하였다. 효능 평가. 6MWT를 기능적 결과 측정으로서 사용하여, 치료전 및 48주에 걸쳐 매 12주 치료 후 이를 실시하였다.
도 3은 에테플러센의 12, 24, 및 48주 후의 디스트로핀-양성 근섬유를 도시한다. 패널 A 및 B는 치료군에 의한 제12, 24, 및 48주에서의 디스트로핀-양성 섬유의 백분율의 기준선으로부터의 평균 절대 변화를 나타낸다. 패널 A에서: *P 값은 제12 및 24주로부터 합한 결과를 사용한 에테플러센 및 위약 간의 비교에 대한 것이고, 고정된 효과로서의 치료 및 공변량으로서 기준선 값 및 DMD 진단 이후의 시간을 이용한 순위 데이터를 위한 공분산 분석 모델에 기초한다. 제시된 평균 변화는 기술 통계학에 기초한 것이다. †P 값은 제48주 값을 기준선과 비교하는 대응표본 t 검정으로부터의 것이다. ‡ 제12 및 24주에 생검된 위약 치료된 환자로부터의 결과가 합해져 있다. § 위약/지연 에테플러센 환자가 제25주에 에테플러센을 투여받기 시작하였고, 제48주에 총 24회 용량을 투여받았다. 약어: BL=기준선; NA=적용가능하지 않음; ND=수행되지 않음; NS=유의미하지 않음; SE=표준 오차.
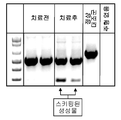
도 4는 디스트로핀-연관 당단백질 착물에 대한 에테플러센의 효과를 제시한다. (4a) 모든 참여 연구 환자에 대한 치료와 관련된 디스트로핀 양성 섬유의 시간-의존성 증가의 대표적인 예. (4b) DMD (a) 및 정상 (c) 대조군 환자 (연구에 속하지 않음), 및 기준선 (b) 및 제48주 (d)에서의 환자 6으로부터의 근육에서의 nNOS μ 염색은 에테플러센에 의한 nNOSμ 결합의 회복을 입증한다. 제48주에 환자 6에서 β-사르코글리칸 (e) 및 γ-사르코글리칸 (f) 염색은 에테플러센에 의한 사르코글리칸 착물의 회복을 입증한다. (4c) RT PCR는 환자 12의 근육에서 치료후 스키핑된 생성물 (289 bp)을 제시한다.
도 5는 에테플러센의 기능적 효능을 그래프로 도시한다. 어두운 자주색 선은 201의 시작으로부터 에테플러센을 투여받은 6명의 평가가능한 환자에 대해 시간에 따른 6MWT 상에서 보행된 거리에서의 기준선으로부터의 변화를 제시한다 (2명의 소년은 제24주 이상에서 불가능했고, 이 분석에서 제외되었다). 회색 선은 처음 24주 동안 위약을 및 마지막 24주 동안 에테플러센을 투여받은 4명의 환자에 대해 6MWT 상에서 보행된 거리에서의 기준선으로부터의 변화를 제시한다.
도 1b는 본 발명의 실시양태에 따른 아르기닌-풍부 펩티드 및 안티센스 올리고머의 접합체를 나타낸다.
도 1c는 골격 연결이 하나 이상의 양으로 하전된 기를 함유하는 도 1b에서와 같은 접합체를 나타낸다.
도 1d 내지 1g는 d 내지 g로 지정된 예시적인 모르폴리노 올리고뉴클레오티드의 반복 서브유닛 절편을 나타낸다.
도 2는 DMD 환자를 치료하는 연구 설계의 개략적 표현이다. 12명의 DMD 환자를 이중-맹검, 위약-대조 연구, 201에서 3개의 코호트 중 하나에 무작위 배정하였다: 코호트 1, 에테플러센 30 mg/kg/wk; 코호트 2, 에테플러센 50 mg/kg/wk; 및 코호트 3, 위약/지연 에테플러센. 제25주에, 코호트 3 내의 위약-치료 환자를 30 또는 50 mg/kg/주 에테플러센을 이용한 개방-표지 치료로 전환하였다. 개방-표지 연장 연구, 202 하에서 환자들을 그들의 동일한 용량의 에테플러센으로 유지시켰다. 근육 생검. 환자는 기준선에서 이두근 생검을 거쳤으며, 디스트로핀의 분석을 위해 제48주에 삼각근 생검을 거쳤다. 제12주에 (코호트 2의 환자 및 코호트 3의 2명의 환자로부터) 또는 제24주에 (코호트 1의 환자 및 코호트 3의 2명의 환자로부터) 추가의 이두근 생검을 수득하였다. 효능 평가. 6MWT를 기능적 결과 측정으로서 사용하여, 치료전 및 48주에 걸쳐 매 12주 치료 후 이를 실시하였다.
도 3은 에테플러센의 12, 24, 및 48주 후의 디스트로핀-양성 근섬유를 도시한다. 패널 A 및 B는 치료군에 의한 제12, 24, 및 48주에서의 디스트로핀-양성 섬유의 백분율의 기준선으로부터의 평균 절대 변화를 나타낸다. 패널 A에서: *P 값은 제12 및 24주로부터 합한 결과를 사용한 에테플러센 및 위약 간의 비교에 대한 것이고, 고정된 효과로서의 치료 및 공변량으로서 기준선 값 및 DMD 진단 이후의 시간을 이용한 순위 데이터를 위한 공분산 분석 모델에 기초한다. 제시된 평균 변화는 기술 통계학에 기초한 것이다. †P 값은 제48주 값을 기준선과 비교하는 대응표본 t 검정으로부터의 것이다. ‡ 제12 및 24주에 생검된 위약 치료된 환자로부터의 결과가 합해져 있다. § 위약/지연 에테플러센 환자가 제25주에 에테플러센을 투여받기 시작하였고, 제48주에 총 24회 용량을 투여받았다. 약어: BL=기준선; NA=적용가능하지 않음; ND=수행되지 않음; NS=유의미하지 않음; SE=표준 오차.
도 4는 디스트로핀-연관 당단백질 착물에 대한 에테플러센의 효과를 제시한다. (4a) 모든 참여 연구 환자에 대한 치료와 관련된 디스트로핀 양성 섬유의 시간-의존성 증가의 대표적인 예. (4b) DMD (a) 및 정상 (c) 대조군 환자 (연구에 속하지 않음), 및 기준선 (b) 및 제48주 (d)에서의 환자 6으로부터의 근육에서의 nNOS μ 염색은 에테플러센에 의한 nNOSμ 결합의 회복을 입증한다. 제48주에 환자 6에서 β-사르코글리칸 (e) 및 γ-사르코글리칸 (f) 염색은 에테플러센에 의한 사르코글리칸 착물의 회복을 입증한다. (4c) RT PCR는 환자 12의 근육에서 치료후 스키핑된 생성물 (289 bp)을 제시한다.
도 5는 에테플러센의 기능적 효능을 그래프로 도시한다. 어두운 자주색 선은 201의 시작으로부터 에테플러센을 투여받은 6명의 평가가능한 환자에 대해 시간에 따른 6MWT 상에서 보행된 거리에서의 기준선으로부터의 변화를 제시한다 (2명의 소년은 제24주 이상에서 불가능했고, 이 분석에서 제외되었다). 회색 선은 처음 24주 동안 위약을 및 마지막 24주 동안 에테플러센을 투여받은 4명의 환자에 대해 6MWT 상에서 보행된 거리에서의 기준선으로부터의 변화를 제시한다.
본 발명의 실시양태는 인간 디스트로핀 유전자에서 엑손 스키핑을 유도하도록 특이적으로 설계된 안티센스 화합물을 투여함으로써 근육 이영양증, 예컨대 DMD 및 BMD를 치료하는 개선된 방법에 관한 것이다. 디스트로핀은 근육 기능에 필수적인 역할을 담당하며, 다양한 근육-관련 질환은 이 유전자의 돌연변이된 형태를 특징으로 한다. 따라서, 특정 실시양태에서, 본원에 기재된 개선된 방법은 DMD 및 BMD에서 발견되는 돌연변이된 디스트로핀 유전자와 같은 인간 디스트로핀 유전자의 돌연변이된 형태에서 엑손 스키핑을 유도하는데 사용될 수 있다.
돌연변이에 의해 유발되는 비정상적 mRNA 스플라이싱 사건으로 인해, 이들 돌연변이된 인간 디스트로핀 유전자는 근육 이영양증의 다양한 형태를 초래하는 상태인 결함성 디스트로핀 단백질을 발현하거나, 측정할 수 있는 디스트로핀을 전혀 발현하지 않는다. 이 상태를 해결하기 위해, 본 발명의 안티센스 화합물은 돌연변이된 인간 디스트로핀 유전자의 예비-프로세싱된 RNA의 선택된 영역에 혼성화하고, 그렇지 않다면 비정상적으로 스플라이싱되는 디스트로핀 mRNA에서 엑손 스키핑 및 차등적 스플라이싱을 유도함으로써, 근육 세포가 기능성 디스트로핀 단백질을 코딩하는 mRNA 전사체를 생성하게 한다. 특정 실시양태에서, 생성된 디스트로핀 단백질은 반드시 디스트로핀의 "야생형" 형태일 필요는 없지만, 다소 디스트로핀의 말단절단된, 여전히 기능성 또는 반-기능성 형태이다.
근육 세포에서 기능성 디스트로핀 단백질의 수준을 증가시킴으로써, 이들 및 관련된 실시양태는 비정상적 mRNA 스플라이싱에 기인한 결함성 디스트로핀 단백질의 발현을 특징으로 하는, 특히 DMD 및 BMD와 같은 근육 이영양증의 형태의 예방 및 치료에 유용하다. 본원에 기재된 방법은 또한 근육 이영양증을 가진 환자를 위한 개선된 치료 선택사항을 제공함으로써, 근육 이영양증의 관련 형태를 치료하는 대안적인 방법에 걸쳐 중요하고 실제적인 이점을 제공한다. 예를 들어, 일부 실시양태에서, 개선된 방법은 이전의 접근법에 비해 보다 높은 용량으로 및/또는 보다 장기간 동안 인간 디스트로핀 유전자에서 엑손 스키핑을 유도하는 안티센스 화합물의 투여에 관한 것이다.
따라서, 본 발명은 환자에서 엑손 스키핑을 유도함으로써 근육 이영양증 예컨대 DMD 및 BMD를 치료하는 개선된 방법에 관한 것이다. 일부 실시양태에서, 엑손 스키핑은, 디스트로핀 프리-mRNA의 엑손 내 표적 서열에 선택적으로 결합하는 전하-중성의 포스포로디아미데이트 모르폴리노 올리고머 (PMO), 예컨대 에테플러센을 포함하는 조성물의 유효량을 투여함으로써 유도된다. 일부 실시양태에서, 본 발명은, 질환을 치료하기에 충분한 일정 기간에 걸쳐 본원에 기재된 안티센스, 예컨대 에테플러센을 포함하는 조성물의 유효량 (예를 들어 적어도 20 mg/kg, 약 25 mg/kg, 약 30 mg/kg 또는 약 30 mg/kg 내지 약 50 mg/kg)으로 DMD 또는 BMD를 치료하는 방법에 관한 것이다.
본 발명의 일부 실시양태는 DMD를 치료하는 질환 조절 요법으로서 에테플러센의 사용에 관한 것이다. 이론에 구애되는 것은 아니지만, 지금까지 에테플러센을 이용하여 관찰된 임상 효능은, 전하-중성의 포스포로디아미데이트 모이어티를 통해 연결된 모르폴린 고리에 결합된 뉴클레오티드를 특징으로 하는 그의 독특한 화학적 조성에 기인하여 그의 안전성 프로파일로부터 도출될 수 있다.
1년 동안 에테플러센으로 치료된 DMD 환자에서, 디스트로핀-양성 섬유의 평균 백분율은 기준선과 비교하여 정상의 47%까지 증가하였다. 증가 정도는 치료 지속기간에 의존적이었다. 디스트로핀 수준의 유의미한 증가는 코호트 1 (30 mg/kg)의 환자로부터 채취된 24-주 생검 및 코호트 3 (제25주에 에테플러센을 시작함)의 환자로부터 채취된 48-주 생검에서 관찰되었다.
에테플러센의 임상 이익은 엑손 스키핑을 유도하여 기능적 디스트로핀 제조를 회복시키는 그의 능력을 반증한다. 임상 효과는 개별 근육 군에서 강도 평가를 넘어서는 지구력 및 근육 용량의 측정인 6MWT로 평가하였다. 시작시부터 30 또는 50 mg/kg 에테플러센을 투여받은 환자는 48주에 걸쳐 안정한 보행 거리를 유지하였는데, 이는 제12주 및 제24주 사이에 신규 디스트로핀 발현에서의 에테플러센-유도된 증가와 일치한다. 이와 대조적으로, 위약/지연 에테플러센 코호트의 환자들은 제36주까지 70 미터를 소실하였으나, 제48주까지 (에테플러센 개시 24주 후) 안정화되는 것으로 나타났다. 이는 연구 시작시부터 30 또는 50 mg/kg 에테플러센을 주 1회 투여받은 환자에서 임상적 영향이 발견된 것과 동일한 기간이다. 두 코호트 모두 하기 기재되는 바와 같이 120주에 걸쳐 안정한 6MWT 결과를 유지하였다.
120주에, 6MWT를 수행할 수 있었던 30 mg/kg 및 50 mg/kg 에테플러센 코호트 내 환자 (변경된 치료 의향이 있는 또는 mITT 집단; n=6)는 보행 능력에 있어 기준선으로부터 13.9 미터, 또는 5% 미만의 약간의 저하를 나타내어 대체적 안정성을 경험하였다. mITT 집단의 경우, 24주의 위약 후 제25주에 치료를 개시한 위약/지연-치료 코호트 (n=4)와 비교하여 64.9 미터의 통계학적으로 유의한 치료 이익 (p ≤0.006)이 관찰되었다. (에테플러센으로의 치료 전) 연구 초기에 상당한 저하를 경험한 후, 위약/지연-치료 코호트도 또한 제36주에서 제120주까지 1.5년 초과 동안 보행 능력에서 안정화를 나타냈으며, 그 기간에 유의미한 수준의 디스트로핀이 마찬가지로 생성되었고, 이 기간에 걸쳐 9.5 미터의 감소가 있었다. 이들 분석은 시험을 이틀 연속 실시하였을 때의 최대 6MWT 점수에 기초하였다.
두 투여 코호트 모두에서 최대 흡기압 및 호기압 (MIP 및 MEP)으로 측정한, 기준선에서 제120주까지 호흡근 기능은 MIP에서 평균 14.6% 증가 및 MEP에서 평균 15.0% 증가를 나타냈다. 예측된 MIP % (체중에 대해 조절된 MIP) 및 예측된 MEP % (연령에 대해 조절된 MEP)는 예측된 MIP %에서 기준선에서의 90.2%에서 제120주에 95.2%로의 평균 증가, 및 예측된 MEP %에서 기준선에서의 79.3%에서 제120주에 79.6%로의 약간의 평균 증가를 나타냈다. 또한, 폐 부피의 척도인 강제 폐활량 (FVC)에서 기준선에서 제120주까지 8.7%의 평균 증가가 있었고, 예측된 FVC % (연령 및 신장에 대해 조절된 FVC)는 제120주까지 평균 90% 초과로 유지되었으며, 기준선에서 101% 및 제120주에서 93%였다.
본 발명은, 적어도 부분적으로, 에테플러센의 치료 효과의 증거에 기초한 것으로, 이는 질환의 기저 원인을 다룸으로써 DMD의 치료에서 중대한 진보를 나타낸다. 따라서, 본 발명은 표적 영역에 특이적으로 혼성화하여 엑손 스키핑을 유도하고 질환을 치료하는, 인간 디스트로핀 유전자의 엑손 내 표적 영역에 상보적인 안티센스 올리고뉴클레오티드, 예컨대 에테플러센을 포함하는 조성물의 유효량을 투여함으로써 환자에서 DMD 또는 BMD를 치료하는 방법에 관한 것이다. 한 실시양태에서, 치료는 치료를 필요로 하는 대상체에게 하나 이상의 본 발명의 안티센스 올리고뉴클레오티드 (예를 들어, 표 3 및 4에 제시된 뉴클레오티드 서열)를, 임의로는 제약 제제 또는 투여 형태의 일부분으로서, 투여함으로써 이루어진다. 치료는 유효량의 하나 이상의 안티센스 올리고뉴클레오티드를 투여함으로써 대상체에서 엑손-스키핑을 유도하는 것을 포함하며, 여기서 엑손은 디스트로핀 유전자로부터의 엑손 1-79 중 임의의 하나 이상이다. 바람직하게는, 엑손은 인간 디스트로핀 유전자로부터의 엑손 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56 또는 8이다.
달리 정의되지 않는다면, 본원에 사용된 모든 기술 및 과학 용어는 본 발명이 속하는 관련 기술분야의 통상의 기술자에 의해 통상적으로 이해되는 것과 동일한 의미를 갖는다. 본원에 기재된 것과 유사하거나 동등한 임의의 방법 및 물질은 본 발명의 실시 또는 시험에 사용될 수 있으며, 바람직한 방법 및 물질이 기재된다. 본 발명의 목적상, 하기 용어는 하기에 정의된다.
I.
정의
"약"은 참조 수량, 수준, 값, 수, 빈도, 퍼센트, 치수, 크기, 양, 중량 또는 길이에 대해 30, 25, 20, 15, 10, 9, 8, 7, 6, 5, 4, 3, 2 또는 1% 만큼 다른 수량, 수준, 값, 수, 빈도, 퍼센트, 치수, 크기, 양, 중량 또는 길이를 의미한다.
용어 "상보적" 및 "상보성"은 염기-쌍형성 규칙에 의해 관련된 폴리뉴클레오티드 (즉, 뉴클레오티드의 서열)를 지칭한다. 예를 들어, 서열 "T-G-A (5'-3')"는 서열 "T-C-A (5'-3')"에 대해 상보적이다. 상보성은 "부분적"일 수 있으며, 여기서 단지 핵산의 염기의 일부만이 염기 쌍형성 규칙에 따라 매칭된다. 또는, 핵산 사이에 "완전한" 또는 "전체적인" 상보성이 있을 수 있다. 핵산 가닥 사이의 상보성의 정도는 핵산 가닥 사이의 혼성화의 효율 및 강도에 유의한 효과를 갖는다. 완벽한 상보성이 때로 바람직한 반면, 일부 실시양태는 표적 RNA에 관해 하나 이상의, 그러나 바람직하게는 6, 5, 4, 3, 2 또는 1개의 미스매치를 포함할 수 있다. 올리고머 내에 임의의 위치에서의 변이가 포함된다. 특정 실시양태에서, 올리고머의 말단 부근의 서열의 변이는 일반적으로 내부의 변이에 대해 바람직하며, 존재할 경우 전형적으로 5' 및/또는 3' 말단의 약 6, 5, 4, 3, 2 또는 1개 뉴클레오티드 내이다.
용어 "세포 관통 펩티드" 및 "CPP"는 상호교환적으로 사용되며, 양이온 세포 관통 페티드를 지칭하고, 또한 수송 펩티드, 담체 펩티드 또는 펩티드 도입 도메인으로도 불린다. 본원에 나타낸 바와 같은 펩티드는 주어진 세포 배양 집단의 세포의 100% 내에서 세포 관통을 유도하는 능력을 가지며, 전신 투여시 생체내에서 다중 조직 내에서 거대분자 전좌를 허용한다. 바람직한 CPP 실시양태는 하기에 추가로 기재된 바와 같은 아르기닌-풍부 펩티드이다.
용어 "안티센스 올리고머" 및 "안티센스 화합물" 및 "안티센스 올리고뉴클레오티드"는 상호교환적으로 사용되며, 염기-쌍형성 모이어티가 왓슨-크릭(Watson-Crick) 염기 쌍형성에 의해 핵산 (전형적으로 RNA)에서 표적 서열에 혼성화하여 표적 서열 내에서 핵산:올리고머 헤테로듀플렉스를 형성하는 것을 허용하는, 서브유닛간 연결에 의해 연결된 염기-쌍형성 모이어티를 각각 갖는 시클릭 서브유닛의 서열을 지칭한다. 시클릭 서브유닛은 리보스 또는 또 다른 펜토스 당 또는 바람직한 실시양태에서 모르폴리노 기 (하기 모르폴리노 올리고머의 설명 참조)에 기초한다. 올리고머는 표적 서열에 대해 정확하거나 근사한 서열 상보성을 가질 수 있으며, 올리고머의 말단 부근의 서열에서의 변이는 일반적으로 내부에서의 변이에 대해 바람직하다.
이러한 안티센스 올리고머는 mRNA의 번역을 차단 또는 억제하도록 또는 천연 프리-mRNA 스플라이스 프로세싱을 억제하도록 설계될 수 있으며, 그것이 혼성화하는 표적 서열"에 대해 지정"되거나 그"에 대해 표적화"되는 것으로 언급될 수 있다. 표적 서열은 전형적으로 mRNA의 AUG 개시 코돈인 번역 억제 올리고머, 또는 예비-프로세싱된 mRNA의 스플라이스 부위인 스플라이스 억제 올리고머 (SSO)를 포함하는 영역이다. 스플라이스 부위에 대한 표적 서열은 예비프로세싱된 mRNA에서 정상적인 스플라이스 수용자 접합의 하류에 그의 5' 말단 1 내지 약 25개 염기쌍을 갖는 mRNA 서열을 포함할 수 있다. 바람직한 표적 서열은 스플라이스 부위를 포함하거나, 엑손 코딩 서열 내에 전체적으로 함유되거나, 스플라이스 수용자 또는 공여자 부위에 걸쳐 있는 예비프로세싱된 mRNA의 임의의 영역이다. 올리고머는 보다 일반적으로 그것이 상기 기재된 방식으로 표적의 핵산에 대해 표적화될 경우, 생물학적으로 관련된 표적, 예컨대 단백질, 바이러스 또는 박테리아"에 대해 표적화"되는 것으로 언급된다.
용어 "모르폴리노 올리고머" 또는 "PMO" (포스포르아미데이트- 또는 포스포로디아미데이트 모르폴리노 올리고머)는 (i) 구조가 1 내지 3개 원자 길이, 바람직하게는 2개 원자 길이, 및 바람직하게는 비하전된 또는 양이온성의, 하나의 서브유닛의 모르폴리노 질소를 인접한 서브유닛의 5' 엑소시클릭 탄소에 결합시키는, 인-함유 연결에 의해 함께 연결되고, (ii) 각각의 모르폴리노 고리가 염기 특이적 수소 결합에 의해 폴리뉴클레오티드의 염기에 결합하는 데 효과적인 퓨린 또는 피리미딘 염기-쌍형성 모이어티를 갖는 모르폴리노 서브유닛 구조로 이루어진 올리고뉴클레오티드 유사체를 지칭한다. 예를 들어, 바람직한 포스포로디아미데이트 연결 유형을 나타내는 도 1a의 구조를 참조한다. 결합 또는 활성을 방해하지 않는 한, 변이가 이 연결에 이루어질 수 있다. 예를 들어, 인에 부착된 산소는 황으로 치환될 수 있다 (티오포스포로디아미데이트). 5' 산소는 아미노 또는 저급 알킬 치환된 아미노로 치환될 수 있다. 인에 부착된 펜던트 질소는 비치환되거나, (임의로 치환된) 저급 알킬로 일치환 또는 이치환될 수 있다. 퓨린 또는 피리미딘 염기 쌍형성 모이어티는 전형적으로 아데닌, 시토신, 구아닌, 우라실, 티민 또는 이노신이다. 모르폴리노 올리고머의 합성, 구조 및 결합 특징은 미국 특허 제5,698,685호, 제5,217,866호, 제5,142,047호, 제5,034,506호, 제5,166,315호, 제5,521,063호, 제5,506,337호, 제8,076,476호, 제8,299,206호 및 제7,943,762호 (양이온성 연결)에 상세화되어 있으며, 이들 모두는 본원에 참조로 포함된다. 변형된 서브유닛간 연결 및 말단 기는 PCT 출원 US2011/038459 및 공보 WO/2011/150408에 상세화되어 있으며, 이들은 그 전문이 본원에 참조로 포함된다.
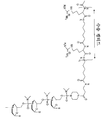
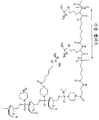
"에테플러센" (또한 "AVN-4658"로도 공지됨)은 염기 서열 5'-CTCCAACATCAAGGAAGATGGCATTTCTAG-3' (서열 1000)을 갖는 PMO이다. 에테플러센은 CAS 등록 번호 1173755-55-9 하에 등록되어 있다. 화학 명칭에는 하기가 포함된다:
및
에테플러센은 다음의 구조를 갖는다:
"아미노산 서브유닛" 또는 "아미노산 잔기"는 α-아미노산 잔기 (-CO-CHR-NH-) 또는 β- 또는 다른 아미노산 잔기 (예를 들어, -CO-(CH2)nCHR-NH-)를 지칭할 수 있으며, 여기서 R은 (수소를 포함할 수 있는) 측쇄이고, n은 1 내지 6, 바람직하게는 1 내지 4이다.
용어 "천연 발생 아미노산"은 자연에서 발견되는 단백질에 존재하는 아미노산을 지칭한다. 용어 "비-천연 아미노산"은 자연에서 발견되는 단백질에 존재하지 않는 아미노산을 지칭하며, 예로는 베타-알라닌 (β-Ala), 6-아미노헥산산 (Ahx) 및 6-아미노펜탄산을 들 수 있다.
"엑손"은 예비-프로세싱된 (또는 전구체) RNA의 모든 부분이 스플라이싱에 의해 제거된 후의, 단백질을 코딩하는 핵산의 한정된 구획, 또는 RNA 분자의 성숙한 형태에서 나타나는 핵산 서열을 지칭한다. 성숙한 RNA 분자는 전령 RNA (mRNA) 또는 rRNA 또는 tRNA와 같은 비-코딩 RNA의 기능성 형태일 수 있다. 인간 디스트로핀 유전자는 약 79개의 엑손을 갖는다.
"인트론"은 단백질로 번역되지 않는 (유전자 내의) 핵산 영역을 지칭한다. 인트론은 전구체 mRNA (프리-mRNA)로 전사되고, 이어서 성숙한 RNA의 형성 동안 스플라이싱에 의해 제거되는 비-코딩 구획이다.
"유효량" 또는 "치료 유효량"은 바람직한 치료 효과를 생성하는 데 유효한 단일 용량으로서 또는 일련의 용량의 부분으로서 인간 대상체에 투여되는, 치료적 화합물, 예컨대 안티센스 올리고뉴클레오티드의 양을 지칭한다. 안티센스 올리고뉴클레오티드에 대해, 이 효과는 전형적으로 선택된 표적 서열의 번역 또는 자연적 스플라이스-프로세싱을 억제함으로써 초래된다. 일부 실시양태에서, 유효량은 대상체를 치료하는 일정 기간 동안 안티센스 올리고뉴클레오티드를 포함하는 적어도 20 mg/kg의 조성물이다. 한 실시양태에서, 유효량은 대상체에서 디스트로핀-양성 섬유의 수를 정상의 적어도 20%까지 증가시키는 안티센스 올리고뉴클레오티드를 포함하는 적어도 20 mg/kg의 조성물이다. 또 다른 실시양태에서, 유효량은 건강한 동년배와 비교하여 환자에서, 예를 들어 6 MWT에서, 20% 결손으로부터의 보행 거리를 안정화, 유지 또는 개선시키는 안티센스 올리고뉴클레오티드를 포함하는 적어도 20 mg/kg의 조성물이다. 또 다른 실시양태에서, 유효량은 적어도 20 mg/kg 내지 약 30 mg/kg, 약 25 mg/kg 내지 약 30 mg/kg, 또는 약 30 mg/kg 내지 약 50 mg/kg이다. 또 다른 실시양태에서, 유효량은 약 30 mg/kg 또는 약 50 mg/kg이다. 또 다른 측면에서, 대상체에서 디스트로핀-양성 섬유의 수를 정상의 적어도 20%, 약 30%, 약 40%, 약 50%, 약 60%, 약 70%, 약 80%, 약 90%, 약 95%까지 증가시키고, 건강한 동년배와 비교하여 환자에서, 예를 들어 6 MWT에서, 20% 결손으로부터의 보행 거리를 안정화, 유지 또는 개선시킬 수 있는 유효량은 적어도 24주, 적어도 36주, 또는 적어도 48주 동안 적어도 20 mg/kg, 약 25 mg/kg, 약 30 mg/kg, 또는 약 30 mg/kg 내지 약 50 mg/kg이다. 한 실시양태에서, 치료는 환자에서 디스트로핀-양성 섬유의 수를 정상의 20-60%, 또는 30-50%까지 증가시킨다.
"엑손 스키핑"은 일반적으로 전체 엑손 또는 그의 부분이 주어진 예비-프로세싱된 RNA로부터 제거됨으로써, 단백질로 번역되는 성숙한 mRNA와 같은 성숙한 RNA에 존재하는 것으로부터 배제되는 프로세스를 지칭한다. 따라서, 그렇지 않다면 스키핑된 엑손에 의해 코딩되는 단백질의 부분은 단백질의 발현된 형태에 존재하지 않으며, 전형적으로 여전히 기능성이지만 변경된 형태의 단백질을 생성한다. 특정 실시양태에서, 스키핑되는 엑손은 인간 디스트로핀 유전자로부터의 비정상적 엑손이며, 이는 그렇지 않다면 비정상적 스플라이싱을 유발하는 그의 서열에 돌연변이 또는 다른 변경을 함유할 수 있다. 특정 실시양태에서, 스키핑되는 엑손은 인간 디스트로핀 유전자의 엑손 1 내지 79 중 임의의 하나 이상, 예컨대 3-8, 10-16, 19-40, 42-47, 및 50-55이지만, 인간 디스트로핀 유전자의 엑손 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56 및 8이 바람직하다.
"디스트로핀"은 막대-형상 세포질 단백질이며, 근육 섬유의 세포골격을 세포막을 통해 주위의 세포외 매트릭스에 연결하는 단백질 복합체의 필수적인 부분이다. 디스트로핀은 다중 기능성 도메인을 함유한다. 예를 들어, 디스트로핀은 약 아미노산 14 내지 240에서 액틴 결합 도메인 및 약 아미노산 253 내지 3040에서 중앙 막대 도메인을 함유한다. 이 큰 중앙 도메인은 약 109개 아미노산의 24개 스펙트린-유사 삼중-나선 요소에 의해 형성되며, 이는 알파-액티닌 및 스펙트린에 대해 상동성을 갖는다. 반복체는 전형적으로 4개의 프롤린-풍부 비-반복 절편에 의해 차단되며, 또한 힌지 영역으로 지칭된다. 반복체 15 및 16은 디스트로핀의 단백질분해성 절단을 위한 주요 부위를 제공하는 것으로 보이는 18 아미노산 스트레치에 의해 분리된다. 대부분의 반복체 사이의 서열 동일성은 10 내지 25% 범위이다. 하나의 반복체는 3개의 알파-이중나선: 1, 2 및 3을 함유한다. 알파-이중나선 1 및 3은 아마도 소수성 계면을 통해 코일화된-코일로서 상호작용하는 7개의 이중나선 턴에 의해 각각 형성된다. 알파-이중나선 2는 보다 복잡한 구조를 가지며, 글리신 또는 프롤린 잔기에 의해 분리되는 4 및 3개의 이중나선 턴의 절편에 의해 형성된다. 각각의 반복체는 2개의 엑손에 의해 코딩되며, 전형적으로 알파-이중나선 2의 제1 부분에서 아미노산 47 및 48 사이의 인트론에 의해 차단된다. 다른 인트론은 반복체에서 상이한 위치에서 발견되며, 통상적으로 이중나선-3에 걸쳐 산포된다. 디스트로핀은 또한 점균류 (딕티오스텔륨 디스코이데움(Dictyostelium discoideum)) 알파-액티닌의 C-말단 도메인에 대해 상동성을 나타내는 시스테인-풍부 절편 (즉, 280개 아미노산 중 15개 시스테인)을 포함하는, 약 아미노산 3080 내지 3360에 시스테인-풍부 도메인을 함유한다. 카르복시-말단 도메인은 약 아미노산 3361 내지 3685이다.
근육속막에서, 디스트로핀의 아미노-말단은 F-액틴에 결합하며, 카르복시-말단은 디스트로핀-연관 단백질 복합체 (DAPC)에 결합한다. DAPC는 디스트로글리칸, 사르코글리칸, 인테그린 및 카베올린을 포함하며, 임의의 이들 성분에서의 돌연변이는 상염색체적으로 유전되는 근육 이영양증을 유발한다. DAPC는 디스트로핀이 부재하는 경우 불안정화되며, 이는 감소된 수준의 막 단백질을 초래하고, 결국 진행성 섬유 손상 및 막 누출을 초래한다. 뒤시엔느 근육 이영양증 (DMD) 및 베커 근육 이영양증 (BMD)과 같은 다양한 형태의 근육 이영양증에서, 부정확한 스플라이싱을 초래하는 유전자 서열에서의 돌연변이에 주로 기인하여 근육 세포는 디스트로핀의 변경되고 기능적으로 결함성 형태를 생성하거나, 전혀 디스트로핀을 생성하지 않는다. 결함성 디스트로핀 단백질의 우세한 발현, 또는 디스트로핀 또는 디스트로핀-유사 단백질의 완전한 소실은 상기 주목된 바와 같이 근육 변성의 급속한 진행을 초래한다. 이와 관련하여, "결함성" 디스트로핀 단백질은 관련 기술분야에서 공지된 바와 같은 DMD 또는 BMD를 갖는 특정 대상체에서 생성되는 디스트로핀의 형태, 또는 검출가능한 디스트로핀의 부재를 특징으로 할 수 있다.
본원에 사용된 바와 같은 용어 "기능" 및 "기능성" 등은 생물학적, 효소적 또는 치료적 기능을 지칭한다.
"기능성" 디스트로핀 단백질은 일반적으로, 전형적으로 DMD 또는 BMD를 갖는 특정 대상체에 존재하는 디스트로핀 단백질의 변경된 또는 "결함성" 형태에 비하여, 그렇지 않다면 근육 이영양증의 특징인 근육 조직의 진행성 분해를 감소시키는 충분한 생물학적 활성을 갖는 디스트로핀 단백질을 지칭한다. 특정 실시양태에서, 기능성 디스트로핀 단백질은 관련 기술분야에서 통상의 기술에 따라 측정된 바와 같은 야생형 디스트로핀의 시험관내 또는 생체내 생물학적 활성의 약 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90% 또는 100% (그 사이의 모든 정수를 포함함)를 가질 수 있다. 한 예로서, 시험관내 근육 배양물에서 디스트로핀-관련된 활성은 근관 크기, 근원섬유 조직화 (또는 해체), 수축성 활성, 및 아세틸콜린 수용체의 자발적 클러스팅에 따라 측정될 수 있다 (예를 들어, 문헌 [Brown et al., Journal of Cell Science. 112:209-216, 1999] 참조). 동물 모델은 또한 질환의 발병기전을 연구하기 위한 가치있는 자원이며, 디스트로핀-관련된 활성을 시험하기 위한 수단을 제공한다. DMD 연구에 가장 폭넓게 사용되는 동물 모델 중 2가지는 mdx 마우스 및 골든 리트리버 근육 이영양증 (GRMD) 개이며, 이들 둘 다는 디스트로핀 음성이다 (예를 들어, 문헌 [Collins & Morgan, Int J Exp Pathol 84: 165-172, 2003] 참조). 이들 및 다른 동물 모델은 다양한 디스트로핀 단백질의 기능적 활성을 측정하는 데 사용될 수 있다. 디스트로핀의 말단절단된 형태, 예컨대 본 발명의 특정 엑손-스키핑 안티센스 화합물에 의해 생성되는 형태가 포함된다.
디스트로핀 합성 또는 제조의 "회복"이라는 용어는 일반적으로, 본원에 기재된 안티센스 올리고뉴클레오티드를 이용한 치료 이후에 근육 이영양증을 가진 환자에서 디스트로핀의 말단절단된 형태를 포함한 디스트로핀 단백질의 제조를 지칭한다. 일부 실시양태에서, 치료는 환자에서 신규 디스트로핀 제조의 1%, 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90% 또는 100% (그 사이의 모든 정수를 포함함) 증가를 가져온다. 일부 실시양태에서, 치료는 대상체에서 디스트로핀-양성 섬유의 수를 정상의 적어도 20%, 약 30%, 약 40%, 약 50%, 약 60%, 약 70%, 약 80%, 약 90 % 또는 약 95% 내지 100%까지 증가시킨다. 다른 실시양태에서, 치료는 대상체에서 디스트로핀-양성 섬유의 수를 정상의 약 20% 내지 약 60%, 또는 약 30% 내지 약 50%까지 증가시킨다. 치료 후의 환자에서 디스트로핀-양성 섬유의 비율은 공지된 기술을 이용한 근육 생검에 의해 결정될 수 있다. 예를 들어, 근육 생검은 환자에서 적합한 근육, 예컨대 상완 이두근 근육으로부터 취해질 수 있다.
양성 디스트로핀 섬유의 백분율에 대한 분석은 치료전 및/또는 치료후 또는 치료 과정 전체 중의 일부 시점에서 실시될 수 있다. 일부 실시양태에서, 치료후 생검은 치료전 생검과는 반대쪽 근육으로부터 취해진다. 치료전 및 치료후 디스트로핀 발현 연구는 디스트로핀에 대한 임의의 적합한 검정을 사용하여 실시될 수 있다. 한 실시양태에서, 디스트로핀에 대한 마커인 항체, 예컨대 모노클로날 또는 폴리클로날 항체를 사용하여 근육 생검으로부터의 조직 절편에 대해 면역조직화학적 검출이 실시된다. 예를 들어, 디스트로핀에 대한 고도로 민감한 마커인 MANDYS106 항체가 사용될 수 있다. 임의의 적합한 2차 항체가 사용될 수 있다.
일부 실시양태에서, % 디스트로핀-양성 섬유는 양성 섬유의 수를 카운팅된 총 섬유로 나눔으로써 계산된다. 정상 근육 샘플은 100% 디스트로핀-양성 섬유를 갖는다. 따라서, % 디스트로핀-양성 섬유는 정상에 대한 백분율로서 표현될 수 있다. 치료전 근육뿐 아니라 복귀 섬유에서 미량의 수준의 디스트로핀의 존재에 대해 통제하기 위해 치료후 근육에서 디스트로핀-양성 섬유를 카운팅할 때 각 환자로부터의 치료전 근육의 절편을 이용하여 기준선을 설정할 수 있다. 이는 상기 환자에서 치료후 근육의 절편에서 디스트로핀-양성 섬유를 카운팅하기 위한 역치로서 사용될 수 있다. 다른 실시양태에서, 항체-염색된 조직 절편은 또한 바이오퀀드(Bioquant) 영상 분석 소프트웨어 (바이오퀀드 이미지 어낼리시스 코포레이션(Bioquant Image Analysis Corporation), 미국 테네시주 내슈빌)를 사용한 디스트로핀 정량화를 위해 사용될 수 있다. 총 디스트로핀 형광 신호 강도는 정상에 대한 백분율로서 기록될 수 있다. 또한, 모노클로날 또는 폴리클로날 항-디스트로핀 항체를 이용한 웨스턴 블롯 분석을 사용하여 디스트로핀 양성 섬유의 백분율을 결정할 수 있다. 예를 들어, 노바카스트라(Novacastra)로부터의 항 디스트로핀 항체 NCL-Dys1을 사용할 수 있다. 디스트로핀-양성 섬유의 백분율은 또한 사르코글리칸 착물의 성분 (β,γ) 및/또는 뉴런 NOS의 발현을 결정함으로써 분석될 수 있다.
일부 실시양태에서, 본 발명의 안티센스 올리고뉴클레오티드, 예컨대 에테플러센을 이용한 치료는 DMD 환자에서 치료 부재시에 예상되었던 진행성 호흡근 기능장애 및/또는 부전을 둔화시키거나 또는 감소시킨다. 한 실시양태에서, 본 발명의 안티센스 올리고뉴클레오티드를 이용한 치료는 치료 부재시에 예상되었던 호흡 보조의 필요성을 감소 또는 제거시킬 수 있다. 한 실시양태에서, 질환의 추세를 추적하는 호흡 기능의 측정, 뿐만 아니라 잠재적 치료적 개입에 대한 평가에는 최대 흡기압 (MIP), 최대 호기압 (MEP) 및 강제 폐활량 (FVC)이 포함된다. MIP 및 MEP는 한 사람이 흡입 및 호기 동안 생성할 수 있는 압력의 수준을 각각 측정하며, 이들은 호흡근 강도에 대한 민감한 척도이다. MIP는 횡경막 근육 약화에 대한 척도이다.
한 실시양태에서, MEP는 MIP 및 FVC를 포함한 다른 폐 기능 검사에서의 변화 이전에 저하될 수 있다. 또 다른 실시양태에서, MEP는 호흡 기능장애에 대한 초기 지시자일 수 있다. 또 다른 실시양태에서, 최대 흡기 후 강제 호기 동안 배출된 공기의 총 부피를 측정하기 위해 FVC가 사용될 수 있다. DMD 환자에서, FVC는 10대 초반까지 신체적 성장에 따라 동반 증가한다. 그러나, 질환 진행에 의해 성장이 둔화 또는 저해되고 근육 약화가 진행됨에 따라, 폐활량은 하향 단계에 진입하여, 10세 내지 12세 이후에는 매년 평균 약 8% 내지 8.5%의 비율로 감소한다. 또 다른 실시양태에서, 예측된 MIP % (체중에 대해 조절된 MIP), 예측된 MEP % (연령에 대해 조절된 MEP) 및 예측된 FVC % (연령 및 신장에 대해 조절된 FVC)는 지지성 분석이다.
"단리된"은 그의 천연 상태에서 정상적으로 이를 수반하는 성분이 실질적으로 또는 본질적으로 없는 물질을 의미한다. 예를 들어, 본원에 사용된 "단리된 폴리뉴클레오티드"는 천연-발생 상태에서 그것에 플랭킹된 서열로부터 정제 또는 제거된 폴리뉴클레오티드, 예를 들어 단편에 정상적으로 인접한 서열로부터 제거된 DNA 단편을 지칭할 수 있다.
본원에 사용된 "충분한 길이"는 표적 디스트로핀 프리-mRNA에서 적어도 8개, 보다 전형적으로 8 내지 30개의 연속적 핵염기에 상보적인 안티센스 올리고뉴클레오티드를 지칭한다. 일부 실시양태에서, 충분한 길이의 안티센스는 표적 디스트로핀 프리-mRNA에서 적어도 8, 9, 10, 11, 12, 13, 14 또는 15개의 연속적 핵염기를 포함한다. 다른 실시양태에서, 충분한 길이의 안티센스는 표적 디스트로핀 프리-mRNA에서 적어도 16, 17, 18, 19, 20, 21, 22, 23, 24 또는 25개의 연속적 핵염기를 포함한다. 충분한 길이의 안티센스 올리고뉴클레오티드는 디스트로핀 유전자의 엑손 1-79 중 임의의 하나 이상에 특이적으로 혼성화할 수 있는 적어도 최소 수의 뉴클레오티드를 갖는다. 바람직하게는, 본 발명의 안티센스 올리고뉴클레오티드는 인간 디스트로핀 유전자의 엑손 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56 또는 8 중 임의의 하나 이상에 특이적으로 혼성화할 수 있는 최소 수의 뉴클레오티드를 갖는다. 바람직하게는, 충분한 길이의 올리고뉴클레오티드는 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39 및 40개 또는 그 초과의 뉴클레오티드의 올리고뉴클레오티드를 포함하는, 길이 약 10 내지 약 50개 뉴클레오티드이다. 한 실시양태에서, 충분한 길이의 올리고뉴클레오티드는 길이 10 내지 약 30개 뉴클레오티드이다. 또 다른 실시양태에서, 충분한 길이의 올리고뉴클레오티드는 길이 15 내지 약 25개 뉴클레오티드이다. 또 다른 실시양태에서, 충분한 길이의 올리고뉴클레오티드는 길이 20 내지 30개, 또는 20 내지 50개 뉴클레오티드이다. 또 다른 실시양태에서, 충분한 길이의 올리고뉴클레오티드는 길이 25 내지 28개 뉴클레오티드이다.
"증진시키다" 또는 "증진시키는", 또는 "증가시키다" 또는 "증가시키는", 또는 "자극하다" 또는 "자극하는"은 일반적으로 안티센스 화합물이 없는 것 또는 대조군 화합물에 의해 유발되는 반응에 비하여, 세포 또는 대상체에서 보다 큰 생리학적 반응 (즉, 하류 효과)를 생성 또는 유발하는 것 또는 안티센스 화합물 또는 조성물의 능력을 지칭한다. 측정가능한 생리학적 반응은 관련 기술분야의 이해 및 본원의 기재로부터 명백한 다른 반응 중에서, 디스트로핀 단백질의 기능성 형태의 증가된 발현, 또는 근육 조직에서의 증가된 디스트로핀-관련된 생물학적 활성을 포함할 수 있다. 약 1%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9%, 10%, 11%, 12%, 13%, 14%, 15%, 16%, 17%, 18%, 19%, 20%, 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95% 또는 100%로 근육 기능의 증가 또는 개선을 포함하는, 증가된 근육 기능이 또한 측정될 수 있다. 약 1%, 2%, %, 15%, 16%, 17%, 18%, 19%, 20%, 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95% 또는 100%의 근육 섬유에서의 증가된 디스트로핀 발현을 포함하는, 기능성 디스트로핀을 발현하는 근육 섬유의 %가 또한 측정될 수 있다. 예를 들어, 섬유의 25 내지 30%가 디스트로핀을 발현할 경우, 근육 기능 개선의 약 40%가 일어날 수 있는 것으로 나타났다 (예를 들어, 문헌 [DelloRusso et al, Proc Natl Acad Sci USA 99: 12979-12984, 2002] 참조). "증가된" 또는 "증진된" 양은 전형적으로 "통계학적으로 유의한" 양이며, 안티센스 화합물이 없는 것 (제제의 부재) 또는 대조군 화합물에 의해 생성되는 양의 1.1, 1.2, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 30, 40, 50배 또는 그 초과 (예를 들어, 500, 1000배) (그 사이 및 1 위의 모든 정수 및 소수점, 예를 들어 1.5, 1.6, 1.7, 1.8 등을 포함함)의 증가를 포함할 수 있다.
"감소시키다" 또는 "억제하다"는 일반적으로 진단 기술분야에서 통상적인 기술에 따라 측정된 바와 같은, 본원에 기재된 질환 또는 상태의 증상과 같은, 관련 생리학적 또는 세포적 반응을 "감소시키는" 본 발명의 하나 이상의 안티센스 화합물의 능력에 관한 것일 수 있다. 관련 생리학적 또는 세포적 반응 (생체내 또는 시험관내)은 관련 기술분야의 통상의 기술자에게 명백할 것이며, DMD 또는 BMD를 갖는 개체에서 발현되는 디스트로핀의 변경된 형태와 같은, 근육 이영양증의 증상 또는 병리의 감소, 또는 디스트로핀의 결함성 형태의 발현의 감소를 포함할 수 있다. 반응의 "감소"는 안티센스 화합물이 없는 것 또는 대조군 조성물에 의해 생성되는 반응에 비하여, 통계학적으로 유의할 수 있으며, 1%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9%, 10%, 11%, 12%, 13%, 14%, 15%, 16%, 17%, 18%, 19%, 20%, 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95% 또는 100% 감소 (그 사이의 모든 정수를 포함함)를 포함할 수 있다.
또한, 본원에 기재된 바와 같은, 표 3 및 4에 나타낸 서열 및 그의 변이체 중 임의의 하나 이상을 포함하는 폴리뉴클레오티드 서열을 발현하는 벡터와 같은, 본 발명의 올리고머성 디스트로핀-표적화 서열을 발현할 수 있는 벡터 전달 시스템이 포함된다. "벡터" 또는 "핵산 구축물"은 예를 들어 폴리뉴클레오티드가 삽입 또는 클로닝될 수 있는 플라스미드, 박테리오파지, 효모 또는 바이러스로부터 유도된 폴리뉴클레오티드 분자, 바람직하게는 DNA 분자를 의미한다. 벡터는 바람직하게는 하나 이상의 고유한 제한 부위를 함유하며, 그의 표적 세포 또는 조직 또는 전구 세포 또는 조직을 포함하는 한정된 숙주 세포에서 자율적 복제를 할 수 있거나, 클로닝된 서열이 재생가능하도록 한정된 숙주의 게놈과 통합될 수 있다. 따라서, 벡터는 자율적으로 복제하는 벡터, 즉 염색체-외 실체로서 존재하는 벡터일 수 있으며, 그의 복제는 염색체 복제, 예를 들어 선형 또는 폐쇄된 원형 플라스미드, 염색체-외 요소, 미니-염색체, 또는 인공 염색체와는 독립적이다. 벡터는 자가-복제를 보증하기 위한 임의의 수단을 함유할 수 있다. 대안적으로, 벡터는 숙주 세포 내로 도입될 경우 게놈 내로 통합되고 그것이 통합되는 염색체(들)과 함께 복제되는 것일 수 있다.
개체 (예를 들어, 포유동물, 예컨대 인간) 또는 세포의 "치료"는 개체 또는 세포의 자연적 과정을 변경하기 위한 시도에 사용되는 임의의 유형의 개입이다. 치료로는 제약 조성물의 투여를 들 수 있으나 이에 제한되지는 않으며, 병리학적 사건의 개시 또는 병인체와의 접촉에 대해 예방적으로 또는 후속으로 수행될 수 있다. 치료는 근육 이영양증의 특정 형태에서와 같이 디스트로핀 단백질과 관련된 질환 또는 상태의 증상 또는 병리에 대한 임의의 바람직한 효과를 포함하며, 예를 들어 치료되는 질환 또는 상태의 하나 이상의 측정가능한 마커에서의 최소의 변화 또는 개선을 포함할 수 있다. 또한, 치료되는 질환 또는 상태의 진행 속도를 감소시키거나, 그 질환 또는 상태의 발병을 지연시키거나, 그의 발병의 중증도를 감소시키는 데 관련될 수 있는 "예방적" 치료가 포함된다. "치료" 또는 "예방"은 반드시 질환 또는 상태, 또는 그의 관련된 증상의 완전한 박멸, 치유 또는 예방을 지시하지는 않는다.
한 실시양태에서, 본 발명의 안티센스 올리고뉴클레오티드를 이용한 치료는 신규 디스트로핀 제조를 증가시키고, 치료 부재시에 예상되었던 보행의 소실을 둔화시키거나 또는 감소시킨다. 예를 들어, 치료는 대상체에서 보행 능력 (예를 들어, 보행의 안정화)을 안정화, 유지, 개선 또는 증가시킬 수 있다. 일부 실시양태에서, 치료는, 예를 들어, 문헌 [McDonald, et al. (Muscle Nerve, 2010; 42:966-74)] (본원에 참조로 포함됨)에 기재된 6분 보행 시험 (6MWT)으로 측정되는 바, 환자에서 안정한 보행 거리를 유지 또는 증가시킨다. 6분 보행 거리 (6MWD)의 변화는 절대값, 백분율 변화 또는 %-예측치의 변화로서 표현될 수 있다. 일부 실시양태에서, 치료는 건강한 동년배와 비교하여 대상체에서 20% 결손으로부터의 6MWT에서의 안정한 보행 거리를 유지 또는 개선시킨다. 건강한 동년배의 전형적인 수행능과 비교한 6MWT에서 DMD 환자의 수행능은 %-예측치를 계산하여 결정될 수 있다. 예를 들어, %-예측 6MWD는 남성의 경우 다음의 방정식을 이용하여 계산할 수 있다: 196.72 + (39.81 x 연령) - (1.36 x 연령2) + (132.28 x 신장 (미터)). 여성의 경우, %-예측 6MWD는 다음의 방정식을 이용하여 계산할 수 있다: 188.61 + (51.50 x 연령) - (1.86 x 연령2) + (86.10 x 신장 (미터)) (본원에 참조로 포함된 문헌 [Henricson et al. PLoS Curr., 2012, 버전 2]). 일부 실시양태에서, 안티센스 올리고뉴클레오티드를 이용한 치료는 환자에서 안정한 보행 거리를 기준선으로부터 3, 5, 6, 7, 8, 9, 10, 15, 20, 25, 30 또는 50 미터 (그 사이의 모든 정수를 포함함) 초과까지 증가시킨다.
DMD 환자에서의 근육 기능의 소실은 정상 소아기 성장 및 발육의 배경을 거술러 일어날 수 있다. 실제로, DMD를 가진 보다 어린 소아는 진행성 근육 장애에도 불구하고 약 1년의 기간에 걸쳐 6MWT 보행 거리의 증가를 보일 수 있다. 일부 실시양태에서, DMD 환자로부터의 6MWD는 전형적으로 발병 대조 대상체 및 연령 및 성별이 일치하는 대상체로부터의 기존 표준 데이터와 비교된다. 일부 실시양태에서, 정상 성장 및 발육은 표준 데이터에 핏팅(fitting)된 연령 및 신장 기반 방정식을 이용하여 확인될 수 있다. 이러한 방정식은 DMD를 가진 대상체에서 6MWD를 퍼센트-예측 (%-예측) 값으로 변환하는데 사용될 수 있다. 특정 실시양태에서, %-예측 6MWD 데이터의 분석은 정상 성장 및 발육을 확인하는 방법을 제시하며, 초기 연령 (예를 들어, 7세 이하)에서의 기능의 획득이 DMD 환자에서의 능력을 개선하는 것이라기 보다는 안정화하는 것을 나타낸다는 것임을 제시할 수 있다 (본원에 참조로 포함된 문헌 [Henricson et al. PLoS Curr., 2012, 버전 2]).
본원에 사용된 "대상체"는 DMD 또는 BMD, 또는 이들 상태와 관련된 임의의 증상 (예를 들어, 근섬유 소실)을 갖거나 가질 위험이 있는 대상체와 같은, 본 발명의 안티센스 화합물로 치료될 수 있는 증상을 나타내거나 증상을 나타낼 위험이 있는 임의의 동물을 포함한다. 적합한 대상체 (환자)는 실험실 동물 (예컨대 마우스, 래트, 토끼 또는 기니아 피그), 농장 동물 및 가축 또는 애완동물 (예컨대 고양이 또는 개)을 포함한다. 비-인간 영장류 및 바람직하게는 인간 환자가 포함된다.
본원에 사용된 "소아 환자"는 1세 내지 21세의 환자 (1세 및 21세 포함)이다.
"알킬" 또는 "알킬렌" 둘 다는 1 내지 18개의 탄소를 함유하는 포화 직쇄 또는 분지쇄 탄화수소 라디칼을 지칭한다. 그 예로는 제한 없이 메틸, 에틸, 프로필, 이소-프로필, 부틸, 이소-부틸, tert-부틸, n-펜틸 및 n-헥실을 들 수 있다. 용어 "저급 알킬"은 1 내지 8개의 탄소를 함유하는 본원에 정의된 바와 같은 알킬기를 지칭한다.
"알케닐"은 2 내지 18개의 탄소를 함유하며, 적어도 하나의 탄소 대 탄소 이중 결합을 포함하는 불포화 직쇄 또는 분지쇄 탄화수소 라디칼을 지칭한다. 그 예로는 제한 없이 에테닐, 프로페닐, 이소-프로페닐, 부테닐, 이소-부테닐, tert-부테닐, n-펜테닐 및 n-헥세닐을 들 수 있다. 용어 "저급 알케닐"은 2 내지 8개의 탄소를 함유하는 본원에 정의된 바와 같은 알케닐기를 지칭한다.
"알키닐"은 2 내지 18개의 탄소를 함유하며, 적어도 하나의 탄소 대 탄소 삼중 결합을 포함하는 불포화 직쇄 또는 분지쇄 탄화수소 라디칼을 지칭한다. 그 예로는 제한 없이 에티닐, 프로피닐, 이소-프로피닐, 부티닐, 이소-부티닐, tert-부티닐, 펜티닐 및 헥시닐을 들 수 있다. 용어 "저급 알키닐"은 2 내지 8개의 탄소를 함유하는 본원에 정의된 바와 같은 알키닐기를 지칭한다.
"시클로알킬"은 모노- 또는 폴리-시클릭 알킬 라디칼을 지칭한다. 그 예로는 제한 없이 시클로부틸, 시클로펜틸, 시클로헥실, 시클로헵틸 및 시클로옥틸을 들 수 있다.
"아릴"은 하나 이상의 폐쇄된 고리(들)를 갖는 내지 18개의 탄소를 함유하는 시클릭 방향족 탄화수소 모이어티를 지칭한다. 그 예로는 제한 없이 페닐, 벤질, 나프틸, 안트라세닐, 페난트라세닐 및 비페닐을 들 수 있다.
"아랄킬"은 화학식 RaRb의 라디칼을 지칭하며, 여기서 Ra는 상기 정의된 바와 같은 알킬렌 쇄이고, Rb는 상기 정의된 바와 같은 하나 이상의 아릴 라디칼, 예를 들어 벤질, 디페닐메틸 등이다.
"티오알콕시"는 화학식 -SRc의 라디칼을 지칭하며, 여기서 Rc는 본원에 정의된 바와 같은 알킬 라디칼이다. 용어 "저급 티오알콕시"는 1 내지 8개의 탄소를 함유하는 본원에 정의된 바와 같은 알콕시기를 지칭한다.
"알콕시"는 화학식 -ORda의 라디칼을 지칭하며, 여기서 Rd는 본원에 정의된 바와 같은 알킬 라디칼이다. 용어 "저급 알콕시"는 1 내지 8개의 탄소를 함유하는 본원에 정의된 바와 같은 알콕시기를 지칭한다. 알콕시기의 예로는 제한 없이 메톡시 및 에톡시를 들 수 있다.
"알콕시알킬"은 알콕시기로 치환된 알킬기를 지칭한다.
"카르보닐"은 C(=O)- 라디칼을 지칭한다.
"구아니디닐"은 H2N(C=NH2)-NH- 라디칼을 지칭한다.
"아미디닐"은 H2N(C=NH2)CH- 라디칼을 지칭한다.
"아미노"는 NH2 라디칼을 지칭한다.
"알킬아미노"는 화학식 -NHRd 또는 -NRdRd의 라디칼을 지칭하며, 여기서 각각의 Rd는 독립적으로 본원에 정의된 바와 같은 알킬 라디칼이다. 용어 "저급 알킬아미노"는 1 내지 8개의 탄소를 함유하는 본원에 정의된 바와 같은 알킬아미노기를 지칭한다.
"헤테로사이클"은 포화, 불포화 또는 방향족이며, 질소, 산소 및 황으로부터 독립적으로 선택되는 1 내지 4개의 헤테로원자를 함유하고, 여기서 질소 및 황 헤테로원자는 임의로 산화될 수 있고, 질소 헤테로원자는 임의로 4급화될 수 있는, 임의의 상기 헤테로사이클이 벤젠 고리에 융합된 비시클릭 고리를 포함하는, 5 내지 7원 모노시클릭, 또는 7 내지 10원 비시클릭 헤테로시클릭 고리를 의미한다. 헤테로사이클은 임의의 헤테로원자 또는 탄소 원자를 통해 부착될 수 있다. 헤테로사이클은 하기 정의되는 바와 같은 헤테로아릴을 포함한다. 따라서, 하기 열거된 헤테로아릴 외에, 헤테로사이클은 또한 모르폴리닐, 피롤리디노닐, 피롤리디닐, 피페리디닐, 피페리지닐, 히단토이닐, 발레로락타밀, 옥시라닐, 옥세타닐, 테트라히드로푸라닐, 테트라히드로피라닐, 테트라히드로피리디닐, 테트라히드로티오페닐, 테트라히드로티오피라닐, 테트라히드로피리미디닐, 테트라히드로티오피라닐 등을 들 수 있다.
"헤테로아릴"은 질소, 산소 및 황으로부터 선택되는 적어도 하나의 헤테로원자를 갖고, 적어도 1개의 탄소 원자를 함유하고, 모노- 및 비시클릭 고리계 둘 다를 포함하는 5 내지 10원의 방향족 헤테로사이클 고리를 의미한다. 대표적인 헤테로아릴은 피리딜, 푸릴, 벤조푸라닐, 티오페닐, 벤조티오페닐, 퀴놀리닐, 피롤릴, 인돌릴, 옥사졸릴, 벤족사졸릴, 이미다졸릴, 벤즈이미다졸릴, 티아졸릴, 벤조티아졸릴, 이속사졸릴, 피라졸릴, 이소티아졸릴, 피리다지닐, 피리미디닐, 피라지닐, 트리아지닐, 신놀리닐, 프탈라지닐 및 퀴나졸리닐이다.
용어 "임의로 치환된 알킬", "임의로 치환된 알케닐", "임의로 치환된 알콕시", "임의로 치환된 티오알콕시", "임의로 치환된 알킬 아미노", "임의로 치환된 저급 알킬", "임의로 치환된 저급 알케닐", "임의로 치환된 저급 알콕시", "임의로 치환된 저급 티오알콕시", "임의로 치환된 저급 알킬 아미노" 및 "임의로 치환된 헤테로시클릴"은 치환될 경우 적어도 하나의 수소 원자가 치환기로 대체되는 것을 의미한다. 옥소 치환기 (=O)의 경우, 2개의 수소 원자가 대체된다. 이와 관련하여, 치환기로는 중수소, 임의로 치환된 알킬, 임의로 치환된 알케닐, 임의로 치환된 알키닐, 임의로 치환된 아릴, 임의로 치환된 헤테로사이클, 임의로 치환된 시클로알킬, 옥소, 할로겐, -CN, -ORx, NRxRy, NRxC(=O)Ry, NRxSO2Ry, -NRxC(=O)NRxRy, C(=O)Rx, C(=O)ORx, C(=O)NRxRy, -SOmRx 및 -SOmNRxRy를 들 수 있으며, 여기서 m은 0, 1 또는 2이고, Rx 및 Ry는 동일하거나 상이하며, 독립적으로 수소, 임의로 치환된 알킬, 임의로 치환된 알케닐, 임의로 치환된 알키닐, 임의로 치환된 아릴, 임의로 치환된 헤테로사이클 또는 임의로 치환된 시클로알킬이고, 각각의 상기 임의로 치환된 알킬, 임의로 치환된 알케닐, 임의로 치환된 알키닐, 임의로 치환된 아릴, 임의로 치환된 헤테로사이클 및 임의로 치환된 시클로알킬 치환기는 하나 이상의 옥소, 할로겐, -CN, -ORx, NRxRy, NRxC(=O)Ry, NRxSO2Ry, -NRxC(=O)NRxRy, C(=O)Rx, C(=O)ORx, C(=O)NRxRy, -SOmRx 및 -SOmNRxRy로 더 치환될 수 있다.
안티센스 분자 명명 시스템은 상이한 안티센스 분자 사이를 구별하기 위해 제안 및 반포되었다 (문헌 [Mann et al., (2002) J Gen Med 4, 644-654] 참조). 이 명명은 몇몇 약간 상이한 안티센스 분자를 시험할 경우에 특히 관련되게 되었으며, 하기 나타낸 바와 같이 모두 동일한 표적 영역에 지정된다:
H#A/D(x:y).
첫번째 문자는 종 (예를 들어 H: 인간, M: 뮤린, C: 개)을 지정한다. "#"은 표적 디스트로핀 엑손 수를 지정한다. "A/D"는 각각 엑손의 시작과 끝에서의 수용자 또는 공여자 스플라이스 부위를 지시한다. (x y)는 "-" 또는 "+"가 각각 인트론 또는 엑손 서열을 지시하는 어닐링 배위자를 나타낸다. 예를 들어, A(-6+18)은 표적 엑손에 선행하는 인트론의 마지막 6개의 염기 및 표적 엑손의 첫번째 18개의 염기를 지시할 것이다. 가장 가까운 스플라이스 부위는 이들 배위자가 "A"에 선행되기 때문에 수용자일 것이다. 공여자 스플라이스 부위에서 어닐링 배위자의 기재는 D(+2-18)일 수 있으며, 여기서 마지막 2개의 엑손 염기 및 첫번째 18개의 인트론 염기는 안티센스 분자의 어닐링 부위에 상응한다. 전체적으로 엑손 어닐링 배위자는 그 엑손의 시작으로부터 65번째와 85번째 뉴클레오티드 사이의 부위인 A(+65+85)로 나타내어질 것이다.
II. 안티센스
올리고뉴클레오티드의 구축
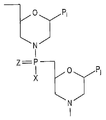
본 발명의 예시적인 실시양태는 도 1a 내지 1c에 도시된 인-함유 골격 연결을 갖는 모르폴리노 올리고뉴클레오티드에 관한 것이다. 바람직한 것은, 본 발명의 한 측면에 따라 그의 골격 연결의 바람직하게는 10% 내지 50%에서 양으로 하전된 기를 함유하도록 변형된 도 1c에 나타낸 바와 같은 포스포로디아미데이트-연결된 모르폴리노 올리고뉴클레오티드이다. 안티센스 올리고머를 포함하는 비하전된 골격 연결을 갖는 모르폴리노 올리고뉴클레오티드는 예를 들어 문헌 [Summerton and Weller 1997] 및 공유된 미국 특허 제5,698,685호, 제5,217,866호, 제5,142,047호, 제5,034,506호, 제5,166,315호, 제5,185,444호, 제5,521,063호, 제5,506,337호, 제8,076,476호, 제8,299,206호 및 제7,943,762호에 상세화되어 있으며, 이들 모두는 명확히 본원에 참조로 포함된다.
모르폴리노-기재 서브유닛의 중요한 특성으로는 1) 안정한 비하전된 또는 양으로 하전된 골격 연결에 의해 올리고머성 형태에 연결되는 능력; 2) 상대적으로 짧은 올리고뉴클레오티드 (예를 들어, 10 내지 15 염기)에서 약 45℃ 위의 Tm 값인 표적 RNA를 포함하는 상보성-염기 표적 핵산과 형성된 중합체가 혼성화할 수 있도록 뉴클레오티드 염기 (예를 들어 아데닌, 시토신, 구아닌, 티미딘, 우라실 및 이노신)를 지지하는 능력; 3) 포유동물 세포 내로 능동적 또는 수동적으로 수송되는 올리고뉴클레오티드의 능력; 및 4) 각각 RNAse 및 RNase H 분해를 저항하는 안티센스 올리고뉴클레오티드:RNA 헤테로듀플렉스의 능력을 들 수 있다.
청구된 대상의 안티센스 올리고뉴클레오티드의 예시적인 골격 구조는 각각 비하전된 또는 양으로 하전된 인-함유 서브유닛 연결에 의해 연결되는 도 1d 내지 1g에 나타낸 모르폴리노 서브유닛 유형을 포함한다. 도 1d는 5개의 원자 반복-단위 골격을 형성하는 인-함유 연결을 나타내며, 여기서 모르폴리노 고리는 1-원자 포스포아미드 연결에 의해 연결된다. 도 1e는 6-원자 반복-단위 골격을 생성하는 연결을 나타낸다. 이 구조에서, 5' 모르폴리노 탄소를 인 기에 연결하는 원자 Y는 황, 질소, 탄소 또는 바람직하게는 산소일 수 있다. 인으로부터의 X 모이어티 펜던트는 모르폴린 또는 피페리딘과 같은 시클릭 구조를 포함하는, 플루오린, 알킬 또는 치환된 알킬, 알콕시 또는 치환된 알콕시, 티오알콕시 또는 치환된 티오알콕시, 또는 비치환된, 1치환된 또는 2치환된 질소일 수 있다. 알킬, 알콕시 및 티오알콕시는 바람직하게는 1 내지 6개의 탄소 원자를 포함한다. Z 모이어티는 황 또는 산소이며, 바람직하게는 산소이다.
도 1f 및 1g에 나타낸 연결은 7-원자 단위-길이 골격을 위해 설계된다. 구조 1f에서, X 모이어티는 구조 1e에서와 같으며, Y 모이어티는 메틸렌, 황, 또는 바람직하게는 산소일 수 있다. 구조 1g에서, X 및 Y 모이어티는 구조 1e에서와 같다. 특히 바람직한 모르폴리노 올리고뉴클레오티드로는 도 1e에 나타낸 형태의 모르폴리노 서브유닛 구조로 이루어진 것을 들 수 있으며, 여기서 X=NH2, N(CH3)2, 또는 1-피페라진 또는 다른 하전된 기이고, Y=O, 및 Z=O이다.
실질적으로 비하전된 올리고뉴클레오티드는 본 발명의 측면에 따라, 하전된 연결, 예를 들어 매 2 내지 5개의 비하전된 연결 당 약 1개 이하, 예컨대 매 10개의 비하전된 연결 당 약 4 내지 5개를 포함하도록 변형될 수 있다. 특정 실시양태에서, 안티센스 활성의 최적 개선은 골격 연결의 약 25%가 양이온성일 경우 나타날 수 있다. 특정 실시양태에서, 소수, 예를 들어 10 내지 20% 양이온성 연결, 또는 양이온성 연결의 수가 50 내지 80% 범위, 예컨대 약 60%로 증진이 나타날 수 있다.
완전히 양이온성-연결된 올리고머를 포함하는 임의의 수의 양이온성 연결을 갖는 올리고머가 제공된다. 그러나, 바람직하게는 올리고머는 예를 들어 10% 내지 80%를 갖는 부분적으로 하전된 것이다. 바람직한 실시양태에서, 약 10% 내지 60%, 바람직하게는 20% 내지 50%의 연결이 양이온성이다.
한 실시양태에서, 양이온성 연결은 골격을 따라 배치된다. 부분적으로 하전된 올리고머는 바람직하게는 적어도 2개의 연속적 비하전된 연결을 함유하며, 즉 올리고머는 바람직하게는 그의 전체 길이를 따라 엄밀하게 교대하는 패턴을 갖지 않는다.
또한, 양이온성 연결의 블록 및 비하전된 연결의 블록을 갖는 올리고머가 고려되며, 예를 들어 비하전된 연결의 중앙 블록은 양이온성 연결의 블록에 의해 플랭킹될 수 있거나, 반대도 마찬가지이다. 한 실시양태에서, 올리고머는 대략 동일-길이 5', 3' 및 중앙 영역을 가지며, 중앙 영역에서 양이온성 연결의 %는 약 50% 초과, 바람직하게는 약 70% 초과이다.
특정 실시양태에서, 안티센스 올리고뉴클레오티드는 혼합물 또는 비하전된 및 양이온성 골격 연결을 갖는 올리고뉴클레오티드의 합성에 관한 상기 및 하기에 인용된 참고문헌, 및 본원의 실시예에서 상세화된 방법을 채용하여, 단계적 고체-상 합성에 의해 제조될 수 있다. 일부의 경우, 예를 들어 약동학을 증진시키거나 화합물의 포획 또는 검출을 용이하게 하기 위해 추가의 화학적 모이어티를 안티센스 화합물에 첨가하는 것이 바람직할 수 있다. 이러한 모이어티는 표준 합성 방법에 따라 공유적으로 부착될 수 있다. 예를 들어, 폴리에틸렌 글리콜 모이어티 또는 다른 친수성 중합체, 예를 들어 1 내지 100개 단량체성 서브유닛을 갖는 것의 첨가가 가용성을 증진시키는 데 유용할 수 있다.
리포터 모이어티, 예컨대 플루오레세인 또는 방사성표지된 기는 검출의 목적으로 부착될 수 있다. 대안적으로, 올리고머에 부착된 리포터 표지는 표지된 항체 또는 스트렙타비딘을 결합할 수 있는 리간드, 예컨대 항원 또는 비오틴일 수 있다. 안티센스 올리고뉴클레오티드의 부착 또는 변형을 위한 모이어티를 선택하는 데 있어서, 일반적으로 물론 생체적합성이며 바람직하지 않은 부작용 없이 대상체에 의해 내성화될 가능성이 있는 기의 화학적 화합물을 선택하는 것이 바람직하다.
안티센스 적용에 사용하기 위한 올리고뉴클레오티드는 일반적으로 길이 약 10 내지 약 50개 서브유닛, 보다 바람직하게는 약 10 내지 30개 서브유닛, 전형적으로 15 내지 25개 염기의 범위이다. 예를 들어, 안티센스 올리고뉴클레오티드에 대한 유용한 길이인 19 내지 20개 서브유닛을 갖는 본 발명의 올리고뉴클레오티드는 이상적으로 2 내지 10개, 예를 들어 4 내지 8개의 양이온성 연결 및 나머지 비하전된 연결을 가질 수 있다. 14 내지 15개 서브유닛을 갖는 올리고뉴클레오티드는 이상적으로 2 내지 7개, 예를 들어 3, 4 또는 5개의 양이온성 연결 및 나머지 비하전된 연결을 가질 수 있다. 바람직한 실시양태에서, 올리고뉴클레오티드는 25 내지 28개 서브유닛을 갖는다.
각각의 모르폴리노 고리 구조는 염기 쌍형성 모이어티를 지지하여, 전형적으로 세포 또는 치료되는 대상체에서 선택된 안티센스 표적에 혼성화하도록 설계된 염기 쌍형성 모이어티의 서열을 형성한다. 염기 쌍형성 모이어티는 천연 DNA 또는 RNA에서 발견되는 퓨린 또는 피리미딘 (예를 들어, A, G, C, T 또는 U) 또는 유사체, 예컨대 히포크산틴 (뉴클레오시드 이노신의 염기 성분) 또는 5-메틸 시토신일 수 있다.
상기 주목된 바와 같이, 특정 실시양태는 PMO-X 올리고머 및 변형된 말단 기를 갖는 것을 포함하는, 신규한 서브유닛간 연결을 포함하는 올리고뉴클레오티드에 관한 것이다. 일부 실시양태에서, 이들 올리고머는 상응하는 비변형된 올리고머가 하는 것보다 DNA 및 RNA에 대해 높은 친화성을 갖고, 다른 서브유닛간 연결을 갖는 올리고머에 비하여 개선된 세포 전달, 효능 및/또는 조직 분포 특성을 발휘한다. 다양한 연결 유형 및 올리고머의 구조적 특징 및 특성은 하기 논의에 보다 상세하게 기재된다. 이들 및 관련된 올리고머의 합성은 공유된 미국 출원 제13/118,298호에 기재되어 있으며, 이는 그 전문이 참고로 도입된다.
특정 실시양태에서, 본 발명은 인간 질환과 관련된 표적 서열에 상보적인 서열을 가지며, 하기 화학식의 뉴클레오티드의 서열을 포함하는 올리고뉴클레오티드 및 그의 제약상 허용되는 염을 제공한다.
여기서, Nu는 핵염기이고;
R1은 화학식
을 갖고,
q는 0, 1 또는 2이고;
R2는 수소, C1-C5 알킬, C1-C5 아랄킬 및 포름아미디닐기로 이루어진 군으로부터 선택되고,
R3은 수소, C1-C10 아실, C1-C10 아미노아실, 천연 또는 비천연 알파 또는 베타 아미노산의 아실 모이어티, C1-C10 아랄킬 및 C1-C10 알킬로 이루어진 군으로부터 선택되거나, 또는
R2 및 R3은 접합되어 5 내지 7원 고리를 형성하며, 여기서 고리는 C1-C10 알킬, 페닐, 할로겐 및 C1-C10 아랄킬로 이루어진 군으로부터 선택되는 치환기로 임의로 치환될 수 있고;
R4는 전자쌍, 수소, C1-C6 알킬 및 C1-C6 아랄킬로 이루어진 군으로부터 선택되고;
Rx는 사르코신아미드, 히드록실, 뉴클레오티드, 세포 관통 펩티드 모이어티 및 피페라지닐로 이루어진 군으로부터 선택되고;
Ry는 수소, C1-C6 알킬, 뉴클레오티드, 세포 관통 펩티드 모이어티, 아미노산, 포름아미디닐기 및 C1-C6 아실로 이루어진 군으로부터 선택되고;
Rz는 전자쌍, 수소, C1-C6 알킬 및 C1-C6 아실로 이루어진 군으로부터 선택된다.
Nu는 아데닌, 구아닌, 티민, 우라실, 시토신 및 히포크산틴으로 이루어진 구으로부터 선택될 수 있다. 보다 바람직하게는, Nu는 티민 또는 우라실이다.
바람직한 실시양태에서, 본 발명은 하기 화학식을 갖는 뉴클레오티드의 서열을 갖는 올리고뉴클레오티드 및 그의 제약상 허용되는 염을 제공한다.
여기서, Nu는 핵염기이고;
R1은 R1' 및 R1"로 이루어진 군으로부터 선택되고, 여기서 R1'는 디메틸-아미노이고, R1"는 화학식
을 갖고,
여기서, 적어도 하나의 R1은 R1"이고;
q는 0, 1 또는 2이되; 단, 적어도 하나의 R1은 피페리디닐 모이어티이고;
R2는 수소, C1-C5 알킬, C1-C5 아랄킬 및 포름아미디닐기로 이루어진 군으로부터 선택되고,
R3은 수소, C1-C10 아실, C1-C10 아미노아실, 천연 또는 비천연 알파 또는 베타 아미노산의 아실 모이어티, C1-C10 아랄킬 및 C1-C10 알킬로 이루어진 군으로부터 선택되거나, 또는
R2 및 R3은 접합되어 5 내지 7원 고리를 형성하며, 여기서 고리는 C1-C10 알킬, 페닐, 할로겐 및 C1-C10 아랄킬로 이루어진 군으로부터 선택되는 치한기로 임의로 치환될 수 있고;
R4는 전자쌍, 수소, C1-C6 알킬 및 아랄킬로 이루어진 군으로부터 선택되고;
Rx는 사르코신아미드, 히드록실, 뉴클레오티드, 세포 관통 펩티드 모이어티 및 피페라지닐로 이루어진 군으로부터 선택되고;
Ry는 수소, C1-C6 알킬, 뉴클레오티드,세포 관통 펩티드 모이어티, 아미노산, 포름아미디닐기 및 C1-C6 아실로 이루어진 군으로부터 선택되고;
Rz는 전자쌍, 수소, C1-C6 알킬 및 C1-C6 아실로 이루어진 군으로부터 선택된다.
Nu는 아데닌, 구아닌, 티민, 우라실, 시토신 및 히포크산틴으로 이루어진 군으로부터 선택될 수 있다. 보다 바람직하게는, Nu는 티민 또는 우라실이다.
R1 기의 약 90 내지 50%는 디메틸아미노 (즉, R1')이다. 보다 바람직하게는, R1 기의 90 내지 50%는 디메틸아미노이다. 가장 바람직하게는, R1 기의 약 66%는 디메틸아미노이다.
R1"는
로 이루어진 군으로부터 선택될 수 있다.
바람직하게는, 올리고뉴클레오티드의 적어도 하나의 뉴클레오티드는 하기 화학식을 갖는다:
여기서, Rx, Ry, Rz 및 Nu는 상기 언급된 바와 같다. 가장 바람직하게는, Nu는 티민 또는 우라실이다.
티민 (T)은 상기 기재된 화학적 변형을 함유하는 바람직한 염기 쌍형성 모이어티 (Nu 또는 Pi)이지만, 관련 기술분야의 통상의 기술자에게 공지된 임의의 염기 서브유닛이 염기 쌍형성 모이어티로서 사용될 수 있다.
올리고뉴클레오티드 및 DNA 또는 RNA는, 각 분자 내의 충분한 수의 상응하는 위치가 서로 수소 결합할 수 있는 뉴클레오티드에 의해 점유될 때 서로 상보적이다. 따라서, "특이적으로 혼성화가능한" 및 "상보적인"은 올리고뉴클레오티드와 DNA 또는 RNA 표적 사이에 안정하고 특이적인 결합이 발생하도록 충분한 정도의 상보성 또는 정확한 쌍형성을 지시하는 데 사용되는 용어이다. 안티센스 분자의 서열은 특이적으로 혼성화가능한 그의 표적 서열의 그것에 100% 상보적일 필요는 없음은 관련 기술분야에서 이해되어 있다. 안티센스 분자는 화합물의 표적 DNA 또는 RNA 분자에의 결합이 표적 DNA 또는 RNA의 정상적인 기능을 방해하여 유용성의 손실을 야기하는 경우 특이적으로 혼성화가능하며, 특이적 결합이 바람직한 조건 하에서, 즉 생체내 검정 또는 치료적 처치의 경우 생리학적 조건 하에서, 및 시험관내 검정의 경우 검정이 수행되는 조건 하에서, 안티센스 화합물의 비-표적 서열에의 비-특이적 결합을 피하기 위한 충분한 정도의 상보성이 있다.
상기 방법이 해당 단백질의 생물학적 기능에 영향없이 단축될 수 있는 단백질 내로부터의 임의의 엑손을 결실시킬 수 있는 안티센스 분자를 선택하는데 사용될 수 있지만, 엑손 결실은 짧아진 전사된 mRNA에서 리딩 프레임 이동을 초래하지 않아야 한다. 따라서, 3개의 엑손의 선형 서열에서 첫번째 엑손의 말단이 코돈에서 3개의 뉴클레오티드 중 2개를 코딩하고, 다음 엑손이 결실될 경우, 선형 서열에서 세번째 엑손은 코돈에 대해 뉴클레오티드 3염기를 완성할 수 있는 단일 뉴클레오티드로 시작해야 한다. 세번째 엑손이 단일 뉴클레오티드로 시작하지 않을 경우, 말단절단된 또는 비-기능성 단백질의 생성을 초래할 리딩 프레임 이동이 있을 것이다.
구조 단백질에서의 엑손의 말단에서 코돈 배열은 항상 코돈의 말단에서 파괴되지는 않을 수 있으며, 결과적으로 mRNA의 프레임-내 리딩을 보장하기 위해 프리-mRNA로부터 하나 초과의 엑손을 결실시킬 필요가 있을 수 있다는 것이 이해될 것이다. 이러한 상황에서, 복수의 안티센스 올리고뉴클레오티드는 각각이 결실되는 엑손에서 스플라이싱을 유도하는 것을 담당하는 상이한 영역에 대해 지정되는 본 발명의 방법에 의해 선택될 필요가 있을 수 있다.
안티센스 분자의 길이는 프리-mRNA 분자 내에서 의도된 위치에 선택적으로 결합할 수 있는 한, 다양할 수 있다. 이러한 서열의 길이는 본원에 기재된 선택 절차에 따라 결정될 수 있다. 일반적으로, 안티센스 분자는 길이 약 10개 뉴클레오티드 내지 길이 약 50개 뉴클레오티드 이하일 것이다. 그러나, 이 범위 내의 임의의 길이의 뉴클레오티드가 본 방법에 사용될 수 있음이 이해될 것이다. 바람직하게는, 안티센스 분자의 길이는 길이 10 내지 30개 뉴클레오티드이다.
안티센스 분자를 생성하는 대부분의 통상의 방법은 2' 히드록시리보스 위치의 메틸화이며, 포스포로티오에이트 골격의 혼입은, 표면적으로는 RNA를 닮았지만 뉴클레아제 분해에 훨씬 더 저항성인 분자를 생성한다.
안티센스 분자로의 이중가닥 형성 동안 프리-mRNA의 분해를 피하기 위해, 상기 방법에서 사용되는 안티센스 분자는 내인성 RNase H에 의한 절단을 최소화 또는 방지하도록 적합화될 수 있다. 이 특성은 세포내 또는 RNase H를 함유하는 조 추출물에서 비메틸화된 올리고뉴클레오티드로의 RNA의 처리가 프리-mRNA: 안티센스 올리고뉴클레오티드 이중가닥의 분해를 초래할 때 매우 바람직하다. 이러한 분해를 우회하거나 유도하지 않을 수 있는 임의의 형태의 변형된 안티센스 분자가 본 방법에 사용될 수 있다. RNA로 이중가닥화될 경우 세포 RNase H에 의해 절단되지 않는 안티센스 분자의 예는 2'-O-메틸 유도체이다. 2'-O-메틸-올리고리보뉴클레오티드는 세포 환경 및 동물 조직에서 매우 안정하며, RNA를 갖는 그의 이중가닥은 그의 리보- 또는 데옥시리보-대응물보다 높은 Tm 값을 갖는다.
RNase H를 활성화시키지 않는 안티센스 분자는 공지된 기술에 따라 제조될 수 있다 (예를 들어 미국 특허 제5,149,797호 참조). 데옥시리보뉴클레오티드 또는 리보뉴클레오티드 서열일 수 있는 이러한 안티센스 분자는 RNase H의 그의 일 원으로서의 올리고뉴클레오티드를 함유하는 이중가닥 분자에의 결합을 입체적으로 장해하거나 방지하는 임의의 구조적 변형을 단순히 함유하며, 여기서 구조적 변형은 이중가닥 형성을 실질적으로 장해 또는 방해하지 않는다. 이중가닥 형성에 관련된 올리고뉴클레오티드의 부분은 그에 대한 RNase H 결합에 관련된 부분과 실질적으로 상이하기 때문에, RNase H를 활성화시키지 않는 많은 안티센스 분자가 이용가능하다. 예를 들어, 이러한 안티센스 분자는 적어도 하나, 또는 전부의 뉴클레오티드-간 브리징 포스페이트 잔기가 변형된 포스페이트, 예컨대 메틸 포스포네이트, 메틸 포스포로티오에이트, 포스포로모르폴리데이트, 포스포로피페라지데이트 및 포스포르아미데이트인 올리고뉴클레오티드일 수 있다. 예를 들어, 뉴클레오티드간 브리징 포스페이트 잔기의 하나 걸러 하나는 기재된 바와 같이 변형될 수 있다. 또 다른 비-제한적 예에서, 이러한 안티센스 분자는 적어도 하나, 또는 전부의 뉴클레오티드가 2' 저급 알킬 모이어티 (예를 들어, C1-C4, 직쇄형 또는 분지형, 포화 또는 불포화 알킬, 예컨대 메틸, 에틸, 에테닐, 프로필, 1-프로페닐, 2-프로페닐 및 이소프로필)을 함유하는 분자이다. 예를 들어, 뉴클레오티드의 하나 걸러 하나는 기재된 바와 같이 변형될 수 있다.
안티센스 올리고뉴클레오티드가 안티센스 분자의 바람직한 형태이지만, 본 발명은, 이에 제한되는 것은 아니나 올리고뉴클레오티드 모방체, 예컨대 하기에 기재되는 것들을 포함한 다른 다른 올리고머성 안티센스 분자를 포괄한다.
본 발명에 유용한 바람직한 안티센스 화합물의 특정 예는 변형된 골격 또는 비-천연 뉴클레오시드간 연결을 함유하는 올리고뉴클레오티드를 포함한다. 본 명세서에서 정의된 바와 같이, 변형된 골격을 갖는 올리고뉴클레오티드는 골격에 인 원자를 보유하는 것 및 골격에 인 원자를 갖지 않는 것을 포함한다. 본 명세서의 목적 상 및 관련 기술분야에서 간혹 언급되는 바와 같이, 그의 뉴클레오시드간 골격에 인 원자를 갖지 않는 변형된 올리고뉴클레오티드는 또한 올리고뉴클레오시드로 간주될 수 있다.
다른 바람직한 올리고뉴클레오티드 모방체에서, 뉴클레오티드 단위의 당 및 뉴클레오시드간 연결, 즉 골격 둘 다는 신규한 기로 대체된다. 염기 단위는 적절한 핵산 표적 화합물과의 혼성화를 위해 유지된다. 우수한 혼성화 특성을 갖는 것으로 나타난 올리고뉴클레오티드 모방체인 하나의 이러한 올리고머성 화합물은 펩티드 핵산 (PNA)으로서 지칭된다. PNA 화합물에서, 올리고뉴클레오티드의 당-골격은 아미드 함유 골격, 특히 아미노에틸글리신 골격으로 대체된다. 핵-염기는 골격의 아미드 부분의 아자 질소 원자에 직접적 또는 간접적으로 보유되고 결합된다.
변형된 올리고뉴클레오티드는 또한 하나 이상의 치환된 당 모이어티를 함유할 수 있다. 올리고뉴클레오티드는 또한 핵염기 (때로 관련 기술분야에서 간단히 "염기"로서 지칭됨) 변형 또는 치환을 포함할 수 있다. 특정 핵-염기는 본 발명의 올리고머성 화합물의 결합 친화성을 증가시키는데 특히 유용하다. 이들로는 2-아미노프로필아데닌, 5-프로피닐우라실 및 5-프로피닐시토신을 포함하는, 5-치환된 피리미딘, 6-아자피리미딘 및 N-2, N-6 및 O-6 치환된 퓨린을 들 수 있다. 5-메틸시토신 치환은 0.6 내지 1.2℃로 핵산 이중가닥 안정성을 증가시키는 것으로 나타났으며, 본원에서 바람직한 염기 치환이고, 2'-O-메톡시에틸 당 변형과 조합시 훨씬 더 특히 그러하다.
본 발명의 올리고뉴클레오티드의 또 다른 변형은 올리고뉴클레오티드의 활성, 세포 분포 또는 세포 흡수를 증진시키는 하나 이상의 모이어티 또는 접합체를 상기 올리고뉴클레오티드에 화학적으로 연결시키는 것을 포함한다. 이러한 모이어티로는 지질 모이어티, 예컨대 콜레스테롤 모이어티, 콜산, 티오에테르, 예를 들어, 헥실-5-트리틸티올, 티오콜레스테롤, 지방족 쇄, 예를 들어, 도데칸디올 또는 운데실 잔기, 인지질, 예를 들어, 디-헥사데실-rac-글리세롤 또는 트리에틸암모늄 1,2-디-O-헥사데실-rac-글리세로-3-H-포스포네이트, 폴리아민 또는 폴리에틸렌 글리콜 쇄, 또는 아다만탄 아세트산, 팔미틸 모이어티, 또는 옥타데실아민 또는 헥실아미노-카르보닐-옥시콜레스테롤 모이어티를 들 수 있으나, 이에 제한되지는 않는다.
주어진 화합물에서 모든 위치는 균일하게 변형될 필요는 없으며, 사실 하나 초과의 상기 언급된 변형은 단일 화합물에서 또는 심지어 올리고뉴클레오티드 내의 단일 뉴클레오시드에서 혼입될 수 있다. 본 발명은 또한 키메라 화합물인 안티센스 화합물을 포함한다. 본 발명의 내용에서, "키메라" 안티센스 화합물 또는 "키메라"는 각각 적어도 하나의 단량체 단위로 이루어진 2개 이상의 화학적으로 분리된 영역, 즉 올리고뉴클레오티드 화합물의 경우 뉴클레오티드를 함유하는 안티센스 분자, 특히 올리고뉴클레오티드이다. 이들 올리고뉴클레오티드는 전형적으로 올리고뉴클레오티드가 뉴클레아제 분해에 대해 증가된 저항성, 증가된 세포 흡수를 제공하도록 변형된 적어도 하나의 영역, 및 표적 핵산에 대한 증가된 결합 친화도를 위한 추가의 영역을 함유한다.
III. 펩티드
수송체
본 발명의 안티센스 화합물은 CPP에 접합된 올리고뉴클레오티드 모이어티, 바람직하게는 세포 내로의 화합물의 수송을 증진시키는 데 효과적인 아르기닌-풍부 펩티드 수송 모이어티를 포함할 수 있다. 수송 모이어티는 바람직하게는 예를 들어 도 1b 및 1c에 나타낸 바와 같이 올리고머의 말단에 부착된다. 펩티드는 주어진 세포 배양 집단의 30%, 40%, 50%, 60%, 70%, 80%, 90% 또는 100%의 세포 (그 사이의 모든 정수를 포함함) 내에서 세포 관통을 유도하는 능력을 가지며, 전신 투여시 생체내에서 다중 조직 내의 거대분자 전좌를 허용한다. 한 실시양태에서, 세포-관통 펩티드는 아르기닌-풍부 펩티드 수송체일 수 있다. 또 다른 실시양태에서, 세포-관통 펩티드는 페네트라틴 또는 Tat 펩티드일 수 있다. 이들 펩티드는 관련 기술분야에 널리 공지되어 있고, 예를 들어 미국 공보 제2010-0016215 A1호에 개시되어 있으며, 이는 그 전문이 참고로 도입된다. 펩티드의 안티센스 올리고뉴클레오티드로의 접합에 대한 특히 바람직한 접근은 PCT 공보 WO2012/150960에서 발견될 수 있으며, 이는 그 전문이 참고로 도입된다. 본 발명의 펩티드 접합된 올리고뉴클레오티드의 바람직한 실시양태는 CPP와 안티센스 올리고뉴클레오티드 사이의 링커로서 글리신을 이용한다. 예를 들어, 바람직한 펩티드 접합된 PMO는 R6-G-PMO로 이루어진다.
상기 기재된 바와 같은 수송 모이어티는 부착된 수송 모이어티의 부재 하에서의 올리고머의 흡수에 비해, 부착된 올리고머의 세포 유입을 크게 증진시키는 것으로 나타났다. 흡수는 바람직하게는 비접합된 화합물에 비해, 적어도 10배, 보다 바람직하게는 20배로 증진된다.
아르기닌-풍부 펩티드 수송체 (즉, 세포-관통 펩티드)의 사용은 본 발명을 실시하는 데 특히 유용하다. 특정 펩티드 수송체는 근육 세포를 포함하는 1차 세포 내로의 안티센스 화합물의 전달에 매우 효과적인 것으로 나타났다. (Marshall, Oda et al. 2007; Jearawiriyapaisarn, Moulton et al. 2008; Wu, Moulton et al. 2008). 더욱이, 다른 공지된 펩티드 수송체, 예컨대 페네트라틴 및 Tat 펩티드에 비하여, 본원에 기재된 펩티드 수송체는 안티센스 PMO에 접합될 경우 몇몇 유전자 전사체의 스플라이싱을 변경하는 증진된 능력을 발휘한다 (Marshall, Oda et al. 2007).
링커를 제외한 예시적인 펩티드 수송체는 하기 표 1에 주어진다.
IV. 제제
및 치료
특정 실시양태에서, 본 발명은 본원에 기재된 바와 같은 안티센스 올리고머의 치료적 전달에 적합한 제제 또는 조성물을 제공한다. 따라서, 특정 실시양태에서, 본 발명은 하나 이상의 제약상 허용되는 담체 (첨가제) 및/또는 희석제와 함께 제제화된, 치료학적-유효량의 하나 이상의 본원에 기재된 올리고머를 포함하는 제약상 허용되는 조성물을 제공한다. 본 발명의 올리고머는 단독으로 투여될 수 있지만, 화합물을 제약 제제 (조성물)로서 투여하는 것이 바람직하다.
본 발명의 조성물은 단독으로 또는 또 다른 치료제와 조합하여 투여될 수 있다. 추가의 치료제는 본 발명의 조성물의 투여 전에, 투여와 동시에 또는 후속적으로 투여될 수 있다. 예를 들어, 조성물은 스테로이드 및/또는 항생제와 조합하여 투여될 수 있다. 스테로이드는 글루코코르티코이드 또는 프레드니손일 수 있다. 글루코코르티코이드, 예컨대 코르티솔은 탄수화물, 지방 및 단백질 대사를 제어하며, 인지질 방출을 방지하고 호산구 작용 및 다수의 다른 메카니즘을 감소시킴으로써 항염증성이다. 미네랄로코르티코이드, 예컨대 알도스테론은, 주로 신장에서 나트륨 저류를 촉진시킴으로써, 전해질 및 물 수준을 제어한다. 코르티코스테로이드는 척추동물의 부신 피질에서 천연적으로 생성되는 스테로이드 호르몬 및 실험실에서 합성한 이들 호르몬의 유사체를 포함한 화학물질의 부류이다. 코르티코스테로이드는, 스트레스 반응, 면역 반응, 및 염증 조절, 탄수화물 대사, 단백질 이화작용, 혈액 전해질 수준, 및 행동을 포함한 넓은 범위의 생리학적 과정에 관여한다. 코르티코스테로이드로는 베타메타손, 부데소니드, 코르티손, 덱사메타손, 히드로코르티손, 메틸프레드니솔론, 프레드니솔론, 및 프레드니손을 들 수 있다.
투여될 수 있는 기타 작용제로는 리아노딘 수용체의 길항제, 예컨대 단트롤렌을 들 수 있는데, 이는 환자 세포 및 DMD의 마우스 모델에서 안티센스-매개 엑손 스키핑을 증진시키는 것으로 나타났다 (본원에 참조로 포함되는 문헌 [G. Kendall et al. Sci Tranl Med 4 164ra160 (2012)]).
핵산 분자의 전달을 위한 방법은 예를 들어 문헌 [Akhtar et al., 1992, Trends Cell Bio., 2:139]; 및 [Delivery Strategies for Antisense Oligonucleotide Therapeutics, ed. Akhtar]; 설리번 등(Sullivan et al.)의 PCT WO 94/02595에 기재되어 있다. 이들 및 다른 프로토콜은 본 발명의 단리된 올리고머를 포함하는, 사실상 임의의 핵산 분자의 전달에 이용될 수 있다.
하기에 상세화된 바와 같이, 본 발명의 제약 조성물은 하기에 적합화된 것들을 포함하여, 고체 또는 액체 형태로 투여하기 위해 특별히 제제화될 수 있다: (1) 경구 투여, 예를 들어 드렌치 (수성 또는 비-수성 용액 또는 현탁액), 정제, 예를 들어 혀로 투여하기 위한 협측, 설하, 및 전신 흡수, 볼루스, 분말, 과립, 페이스트에 대해 표적화된 것; (2) 예를 들어 멸균 용액 또는 현탁액, 또는 지속-방출 제제로서의, 예를 들어 피하, 근육내, 정맥내 또는 경막외 주사에 의한 비경구 투여; (3) 피부에 도포되는 크림, 연고, 또는 조절-방출 패치 또는 스프레이로서의 국소 적용; (4) 예를 들어 질좌제, 크림 또는 폼으로서의 질내 또는 직장내; (5) 설하; (6) 안구; (7) 경피; 또는 (8) 비강.
어구 "제약상 허용되는"은 본원에서 건전한 의학적 판단의 범위 내에서 합리적인 이익/위험 비율과 상응하는 과도한 독성, 자극, 알러지 반응, 또는 다른 문제 또는 합병증 없이 인간 및 동물의 조직에 접촉하여 사용하기에 적합한 화합물, 물질, 조성물 및/또는 투여 형태를 지칭하기 위해 채용된다.
본원에 사용된 어구 "제약상 허용되는 담체"는 대상 화합물을 하나의 기관 또는 신체의 부분으로부터 또 다른 기관 또는 신체의 부분으로 전달 또는 수송하는 데 관련되는, 제약상 허용되는 물질, 조성물 또는 비히클, 예컨대 액체 또는 고체 충전제, 희석제, 부형제, 제조 보조제 (예를 들어, 윤활제, 탈크 마그네슘, 칼슘 또는 아연 스테아레이트, 또는 스테아르산), 또는 용매 캡슐화 물질을 의미한다. 각각의 담체는 제제의 다른 성분과의 혼화성이고, 환자에게 손상을 주지 않는 의미에서 "허용되어야" 한다.
제약상 허용되는 담체로서 기능할 수 있는 물질의 일부 예로는, 제한 없이 (1) 당, 예컨대 락토스, 글루코스 및 수크로스; (2) 전분, 예컨대 옥수수 전분 및 감자 전분; (3) 셀룰로스 및 그의 유도체, 예컨대 나트륨 카르복시메틸 셀룰로스, 에틸 셀룰로스 및 셀룰로스 아세테이트; (4) 분말화된 트라가칸트; (5) 맥아; (6) 젤라틴; (7) 탈크; (8) 부형제, 예컨대 코코아 버터 및 좌제 왁스; (9) 오일, 예컨대 땅콩유, 면실유, 해바라기유, 참깨유, 올리브유, 옥수수유 및 대두유; (10) 글리콜, 예컨대 프로필렌 글리콜; (11) 폴리올, 예컨대 글리세린, 소르비톨, 만니톨 및 폴리에틸렌 글리콜; (12) 에스테르, 예컨대 에틸 올레에이트 및 에틸 라우레이트; (13) 한천; (14) 완충제, 예컨대 수산화마그네슘 및 수산화알루미늄; (15) 알긴산; (16) 피로겐-무함유 수; (17) 등장성 염수; (18) 링거 용액; (19) 에틸 알코올; (20) pH 완충된 용액; (21) 폴리에스테르, 폴리카르보네이트 및/또는 폴리무수물; 및 (22) 제약 제제에 채용되는 다른 비-독성 혼화성 물질을 들 수 있다.
본 발명의 안티센스 올리고머와 제제화하기에 적합한 제제의 추가의 비제한적 예로는 다양한 조직 내로의 약물의 유입을 증진시킬 수 있는 PEG 접합된 핵산, 인지질 접합된 핵산, 친지질성 모이어티를 함유하는 핵산, 포스포로티오에이트, P-당단백질 억제제 (예컨대 프루로닉 P85); 이식 후의 지속 방출 전달을 위한 생체분해성 중합체, 예컨대 폴리 (DL-락티드-코글리콜리드) 미소구 (Emerich, D F et al., 1999, Cell Transplant, 8, 47-58) (메사추세츠주 캠브리지 소재 알컴즈 인코포레이티드 (Alkermes, Inc.)); 및 혈액 뇌 장벽을 통해 약물을 전달할 수 있으며, 신경 흡수 메커니즘을 변경시킬 수 있는 로딩된 나노입자, 에컨대 폴리부틸시아노아크릴레이트로 제조된 것 (Prog Neuropsychopharmacol Biol Psychiatry, 23, 941-949, 1999)을 들 수 있다.
본 발명은 또한 폴리 (에틸렌 글리콜) 지질 (PEG-변형된, 분지된 및 비분지된 또는 그의 조합, 또는 오랜 시간-순환하는 리포솜 또는 잠행성 리포솜)을 함유하는 표면-변형된 리포솜을 포함하는 조성물의 용도를 특징으로 한다. 본 발명의 올리고머는 또한 다양한 분자량의 공유적으로 부착된 PEG 분자를 포함한다. 이들 제제는 표적 조직에서의 약물의 축적을 증가시키는 방법을 제공한다. 이 부류의 약물 담체는 단핵 포식세포계 (MPS 또는 RES)에 의해 옵소닌화 및 배설에 저항함으로써, 캡슐화된 약물에 대한 보다 오랜 혈액 순환 시간 및 증진된 조직 노출을 가능하게 한다 (Lasic et al. Chem. Rev. 1995, 95, 2601-2627; Ishiwata et al., Chem. Pharm. Bull. 1995, 43, 1005-1011). 이러한 리포솜은 아마도 분출에 의해 종양에서 선택적으로 축적되며, 혈관신생된 표적 조직에서 포획되는 것으로 나타났다 (Lasic et al., Science 1995, 267, 1275-1276; Oku et al., 1995, Biochim. Biophys. Acta, 1238, 86-90). 오랜 시간-순환하는 리포솜은 특히 MPS의 조직에서 축적되는 것으로 공지된 통상적인 양이온성 리포솜에 비하여, DNA 및 RNA의 약동학 및 약역학을 증진시킨다 (Liu et al., J. Biol. Chem. 1995, 42, 24864-24870; 최 등 (Choi et al.), 국제 PCT 공보 제WO 96/10391호; 안셀 등 (Ansell et al.), 국제 PCT 공보 제WO 96/10390호; 홀란드 등 (Holland et al.), 국제 PCT 공보 제WO 96/10392호). 오랜 시간-순환하는 리포솜은 또한 간 및 비장과 같은 물질대사적으로 공격적인 MPS 조직에서의 축적을 피하는 그들의 능력에 기초하여, 양이온성 리포솜에 비하여 보다 큰 정도로 뉴클레아제 분해로부터 약물을 보호할 가능성이 있다.
추가의 실시양태에서, 본 발명은 미국 특허 제6,692,911호, 제7,163,695호 및 제7,070,807호에 기재된 바와 같은 전달을 위해 제조된 올리고머 조성물을 포함한다. 이와 관련하여, 한 실시양태에서, 본 발명은 리신 및 히스티딘 (HK)의 공중합체를 포함하는 조성물 (미국 특허 제7,163,695호, 제7,070,807호 및 제6,692,911호에 기재된 바와 같음)에서의, 단독으로 또는 PEG (예를 들어, 분지된 또는 비분지된 PEG 또는 둘 다의 혼합물)과 조합으로, PEG 및 표적화 모이어티 또는 가교제와 조합으로의 임의의 상기의 것과 조합으로 본 발명의 올리고머를 제공한다. 특정 실시양태에서, 본 발명은 글루콘산-변형된 폴리히스티딘 또는 글루코닐화-폴리히스티딘/트랜스페린-폴리리신을 포함하는 조성물에서의 안티센스 올리고머를 제공한다. 관련 기술분야의 통상의 기술자는 또한 His 및 Lys와 유사한 특성을 갖는 아미노산이 조성물 내에서 치환될 수 있음을 인지할 것이다.
본원에 기재된 올리고머의 특정 실시양태는 염기성 관능기, 예컨대 아미노 또는 알킬아미노를 함유할 수 있으며, 따라서 제약상 허용되는 산과 함께 제약상 허용되는 염을 형성할 수 있다. 이와 관련하여 용어 "제약상 허용되는 염"은 본 발명의 화합물의 상대적으로 비-독성, 무기 및 유기 산 부가염을 지칭한다. 이들 염은 계내에서 투여 비히클 또는 투여 형태 제조 공정으로, 또는 본 발명의 정제된 화합물을 그의 유리 염기 형태로 적합한 유기 또는 무기산과 별개로 반응시키고, 후속 정제 동안 이렇게 형성되는 염을 단리함으로써 제조될 수 있다. 대표적인 염으로는 히드로브로마이드, 히드로클로라이드, 술페이트, 비술페이트, 포스페이트, 니트레이트, 아세테이트, 발레레이트, 올레에이트, 팔미테이트, 스테아레이트, 라우레이트, 벤조에이트, 락테이트, 포스페이트, 토실레이트, 시트레이트, 말레에이트, 푸마레이트, 숙시네이트, 타르트레이트, 나프틸레이트, 메실레이트, 글루코헵토네이트, 락토비오네이트 및 라우릴술포네이트 염 등을 들 수 있다 (예를 들어, 문헌 [Berge et al. (1977) "Pharmaceutical Salts", J. Pharm. Sci. 66:1-19] 참조).
대상 올리고머의 제약상 허용되는 염은 예를 들어 비-독성 유기 또는 무기산으로부터의 화합물의 통상적인 비독성 염 또는 4급 암모늄염을 포함한다. 예를 들어, 이러한 통상적인 비독성 염으로는 무기산, 예컨대 히드로클로라이드, 브롬화수소산, 황산, 술팜산, 인산, 질산 등으로부터 유도된 것; 및 유기산, 예컨대 아세트산, 프로피온산, 숙신산, 글리콜산, 스테아르산, 락트산, 말산, 타르타르산, 시트르산, 아스코르브산, 팔미트산, 말레산, 히드록시말레산, 페닐아세트산, 글루탐산, 벤조산, 살리시클릭산, 술파닐산, 2-아세톡시벤조산, 푸마르산, 톨루엔술폰산, 메탄술폰산, 에탄 디술폰산, 옥살산, 이소티온산 등으로부터 제조된 염을 들 수 있다.
특정 실시양태에서, 본 발명의 올리고머는 하나 이상의 산성 관능기를 함유할 수 있으며, 따라서 제약상 허용되는 염기와 함께 제약상 허용되는 염을 형성할 수 있다. 이들 예에서 용어 "제약상 허용되는 염"은 본 발명의 화합물의 상대적으로 비-독성, 무기 및 유기 염기 부가염을 지칭한다. 이들 염은 유사하게 계내에서 투여 비히클 또는 투여 형태 제조 공정으로, 또는 정제된 화합물을 그의 유리 산 형태로 적합한 염기, 예컨대 제약상 허용되는 금속 양이온의 히드록시드, 카르보네이트 또는 비카르보네이트와, 암모니아와, 또는 제약상 허용되는 유기 1차, 2차 또는 3차 아민과 별개로 반응시킴으로써 제조될 수 있다. 대표적인 알칼리 또는 알칼리토 염으로는 리튬, 나트륨, 칼륨, 칼슘, 마그네슘 및 알루미늄 염 등을 들 수 있다. 염기 부가염의 형성에 유용한 대표적인 유기 아민으로는 에틸아민, 디에틸아민, 에틸렌디아민, 에탄올아민, 디에탄올아민, 피페라진 등을 들 수 있다 (예를 들어, 버지 등 (Berge et al.), 상기 문헌 참조).
습윤제, 유화제 및 윤활제, 예컨대 나트륨 라우릴 술페이트 및 마그네슘 스테아레이트, 및 착색제, 이형제, 코팅제, 감미제, 향미제 및 방향제, 보존제 및 항산화제는 또한 조성물에 존재할 수 있다.
제약상 허용되는 항산화제의 예로는 (1) 수용성 항산화제, 예컨대 아스코르브산, 시스테인 히드로클로라이드, 나트륨 비술페이트, 나트륨 메타비술파이트, 나트륨 술파이트 등; (2) 지용성 항산화제, 예컨대 아스코르빌 팔미테이트, 부틸화 히드록시아니솔 (BHA), 부틸화 히드록시톨루엔 (BHT), 레시틴, 프로필 갈레이트, 알파-토코페롤 등; 및 (3) 금속 킬레이트제, 예컨대 시트르산, 에틸렌디아민 테트라아세트산 (EDTA), 소르비톨, 타르타르산, 인산 등을 들 수 있다.
본 발명의 제제는 경구, 비강, 국소 (협측 및 설하를 포함함), 직장, 질내 및/또는 비경구 투여에 적합한 것을 포함한다. 제제는 편리하게는 단위 투여 형태로 제시될 수 있으며, 약학 기술분야에서 널리 공지된 임의의 방법에 의해 제조될 수 있다. 단일 투여 형태를 제조하기 위한 담체 물질과 조합될 수 있는 활성 성분의 양은 치료되는 숙주, 특정 투여 방식에 따라 다양할 것이다. 단일 투여 형태를 제조하기 위한 담체 물질과 조합될 수 있는 활성 성분의 양은 일반적으로 치료 효과를 생성하는 화합물의 양일 것이다. 일반적으로, 100% 중, 이 양은 활성 성분의 약 0.1% 내지 약 99%, 바람직하게는 약 5% 내지 약 70%, 가장 바람직하게는 약 10% 내지 약 30%의 범위일 것이다.
특정 실시양태에서, 본 발명의 제제는 시클로덱스트린, 셀룰로스, 리포솜, 미셀 형성제, 예를 들어 담즙산, 및 중합체성 담체, 예를 들어 폴리에스테르 및 폴리무수물; 및 본 발명의 올리고머를 포함한다. 특정 실시양태에서, 상기언급된 제제는 본 발명의 올리고머를 경구적으로 생체이용가능하게 만든다.
이들 제제 또는 조성물의 제조 방법은 본 발명의 올리고머를 담체, 및 임의로 하나 이상의 부속 성분과 회합시키는 단계를 포함한다. 일반적으로, 제제는 본 발명의 화합물을 액체 담체, 또는 미세하게 분리된 고체 담체, 또는 둘 다와 균일하고 긴밀하게 회합시킨 후, 필요할 경우 생성물을 성형함으로써 제조된다.
경구 투여에 적합한 본 발명의 제제는 캡슐제, 카세제, 환제, 정제, 로젠지제 (향미된 베이시스, 통상적으로 수크로스 및 아카시아 또는 트라가칸트를 사용함), 분말제, 과립제의 형태로, 또는 수성 또는 비-수성 액체 중의 용액 또는 현택액으로서, 또는 수중유 또는 유중수 액체 에멀젼으로서, 또는 엘릭시르제 또는 시럽제로서, 또는 캔디제 (불활성 염기, 예컨대 젤라틴 및 글리세린, 또는 수크로스 및 아카시아를 사용함)로서, 및/또는 구강 청결제로서 등일 수 있으며, 이들 각각은 미리 결정된 양의 본 발명의 화합물을 활성 성분으로서 함유한다. 본 발명의 올리고머는 또한 볼루스제, 연약 또는 페이스트제로서 투여될 수 있다.
경구 투여를 위한 본 발명의 고체 투여 형태 (캡슐제, 정제, 환제, 당제, 분말제, 과립제, 트로키제 등)에서, 활성 성분은 하나 이상의 제약상 허용되는 담체, 예컨대 나트륨 시트레이트 또는 디칼슘 포스페이트, 및/또는 하기 중 임의의 것과 혼합될 수 있다: (1) 충전제 또는 증량제, 예컨대 전분, 락토스, 수크로스, 글루코스, 만니톨 및/또는 규산; (2) 결합제, 예를 들어 카르복시메틸셀룰로스, 알기네이트, 젤라틴, 폴리비닐 피롤리돈, 수크로스 및/또는 아카시아; (3) 보습제, 예컨대 글리세롤; (4) 붕괴제, 예컨대 한천-한천, 탄산칼슘, 감자 또는 타피오카 전분, 알긴산, 특정 실리케이트 및 탄산나트륨; (5) 용액 지연제, 예컨대 파라핀; (6) 흡수 촉진제, 예컨대 4급 암모늄 화합물 및 계면활성제, 예컨대 폴록사머 및 나트륨 라우릴 술페이트; (7) 습윤제, 예를 들어 세틸 알코올, 글리세롤 모노스테아레이트, 및 비-이온성 계면활성제; (8) 흡수제, 예컨대 카올린 및 벤토나이트 점토; (9) 윤활제, 예컨대 탈크, 칼슘 스테아레이트, 마그네슘 스테아레이트, 고체 폴리에틸렌 글리콜, 나트륨 라우릴 술페이트, 아연 스테아레이트, 나트륨 스테아레이트, 스테아르산, 및 이들의 혼합물; (10) 착색제; 및 (11) 제어 방출제, 예컨대 크로스포비돈 또는 에틸 셀룰로스. 캡슐제, 정제 및 환제의 경우, 제약 조성물은 또한 완충제를 포함할 수 있다. 유사한 유형의 고체 조성물은 또한 부형제, 예컨대 락토스 또는 유당, 및 고분자량 폴리에틸렌 글리콜 등을 사용한 연질 및 경질-쉘화된 젤라틴 캡슐 중의 충전제로서 채용될 수 있다.
정제는 임의로 하나 이상의 부속 성분과 함께 압축 또는 성형함으로써 제조될 수 있다. 압축된 정제는 결합제 (예를 들어, 젤라틴 또는 히드록시프로필메틸 셀룰로스), 윤활제, 불활성 희석제, 보존제, 붕괴제 (예를 들어, 나트륨 전분 글리콜레이트 또는 가교된 나트륨 카르복시메틸 셀룰로스), 표면-활성제 또는 분산제를 사용하여 제조될 수 있다. 성형된 정제는 불활성 액체 희석제로 습윤화된 분말화된 화합물의 혼합물을 적합한 기계에서 성형함으로써 제조될 수 있다.
본 발명의 제약 조성물의 정제, 및 다른 고체 투여 형태, 예컨대 당제, 캡슐제, 환제 및 과립제는 임의로 코팅 및 쉘, 예컨대 장용성 코팅 및 제약-제제화 기술분야에서 널리 공지된 다른 코팅으로 스코어링 또는 제조될 수 있다. 이들은 또한 예를 들어 바람직한 방출 프로파일을 제공하는 다양한 비율의 히드록시프로필메틸 셀룰로스, 다른 중합체 매트릭스, 리포솜 및/또는 미소구를 사용하여 그 안의 활성 성분의 저속 또는 제어된 방출을 제공하도록 제제화될 수 있다. 이들은 급속 방출을 위해 제제화, 예를 들어 동결-건조될 수 있다. 이들은 예를 들어 박테리아-보유 필터를 통한 여과에 의해, 또는 사용 직전에 멸균수, 또는 일부 다른 멸균 주사가능한 매질에 용해될 수 있는 멸균 고체 조성물의 형태로 멸균제를 혼입함으로써 멸균될 수 있다. 이들 조성물은 또한 임의로 불투명화제를 함유할 수 있으며, 활성 성분(들)만을, 또는 우선적으로 위장관의 특정 부분에서, 임의로 지연된 방식으로 방출하는 조성물일 수 있다. 사용될 수 있는 매립 조성물의 예로는 중합체성 물질 및 왁스를 들 수 있다. 활성 성분은 또한 적절할 경우 하나 이상의 상기 기재된 부형제를 갖는 미소-캡슐화된 형태일 수 있다.
본 발명의 화합물의 경구 투여를 위한 액체 투여 형태는 제약상 허용되는 에멀젼, 마이크로에멀젼, 용액, 현탁액, 시럽 및 엘릭시르를 포함한다. 활성 성분 외에, 액체 투여 형태는 관련 기술분야에서 통상적으로 사용되는 불활성 희석제, 예를 들어 물 또는 다른 용매, 가용화제 및 유화제, 예컨대 에틸 알코올, 이소프로필 알코올, 에틸 카르보네이트, 에틸 아세테이트, 벤질 알코올, 벤질 벤조에이트, 프로필렌 글리콜, 1,3-부틸렌 글리콜, 오일 (특히, 면실유, 땅콩유, 옥수수유, 발아유, 올리브유, 피마자유 및 참깨유), 글리세롤, 테트라히드로푸릴 알코올, 폴리에틸렌 글리콜 및 소르비탄의 지방산 에스테르, 및 이들의 혼합물을 함유할 수 있다.
불활성 희석제 외에, 경구 조성물은 또한 아쥬반트, 예컨대 습윤제, 유화제 및 현탁화제, 감미제, 향미제, 착색제, 방향제 및 보존제를 포함할 수 있다.
활성 화합물 외에 현탁액은 현탁화제, 예를 들어 에톡실화 이소스테아릴 알코올, 폴리옥시에틸렌 소르비톨 및 소르비탄 에스테르, 미세결정질 셀룰로스, 알루미늄 메타히드록시드, 벤토나이트, 한천-한천 및 트라가칸트, 및 이들의 혼합물을 함유할 수 있다.
직장 또는 질내 투여를 위한 제제는 하나 이상의 본 발명의 화합물을 예를 들어 코코아 버터, 폴리에틸렌 글리콜, 좌제 왁스 또는 살리실레이트를 포함하는 하나 이상의 적합한 비자극성 부형제 또는 담체와 혼합함으로써 제조될 수 있고, 실온에서 고체이지만, 체온에서는 액체이고, 따라서 직장 또는 질강에서 용해되어 활성 화합물을 방출하는 좌제로서 제시될 수 있다.
본원에서 제공된 바와 같은 올리고머의 국소 또는 경피 투여를 위한 제제 또는 투여 형태는 분말제, 스프레이제, 연고, 페이스트, 크림, 로션, 겔, 용액, 패치 및 흡입제를 포함한다. 활성 올리고머는 멸균 상태 하에서 제약상 허용되는 담체, 및 필요할 수 있는 임의의 보존제, 완충제 또는 추진제와 혼합될 수 있다. 연고, 페이스트, 크림 및 겔은 본 발명의 활성 화합물 외에 부형제, 예컨대 동물성 및 식물성 지방, 오일, 왁스, 파라핀, 전분, 트라가칸트, 셀룰로스 유도체, 폴리에틸렌 글리콜, 실리콘, 벤토나이트, 규산, 탈크 및 산화아연, 또는 이들의 혼합물을 함유할 수 있다.
분말제 및 스프레이제는 본 발명의 올리고머 외에 부형제, 예컨대 락토스, 탈크, 규산, 수산화알루미늄, 칼슘 실리케이트 및 폴리아미드 분말, 또는 이들 물질의 혼합물을 함유할 수 있다. 스프레이제는 통상적인 추진제, 예컨대 클로로플루오로히드로카본 및 휘발성 비치환된 탄화수소, 예컨대 부탄 및 프로판을 추가로 함유할 수 있다.
경피 패치는 본 발명의 올리고머의 신체로의 제어된 전달을 제공하는 추가의 이점을 갖는다. 이러한 투여 형태는 올리고머를 적절한 매질에 용해 또는 분산시킴으로써 제조될 수 있다. 흡수 증진제는 또한 피부를 통한 제제의 유동을 증가시키는 데 사용될 수 있다. 이러한 유동의 속도는 관련 기술분야에서 공지된 다른 방법 중에서 속도 제어 막을 제공하거나, 제제를 중합체 매트릭스 또는 겔에 분산시킴으로써 제어될 수 있다.
비경구 투여에 적합한 제약 조성물은 하나 이상의 본 발명의 올리고머를 의도되는 수용자의 혈액과 제제를 등장성이 되도록 하는, 당, 알코올, 항산화제, 완충제, 정균제, 용질 또는 현탁화제 또는 증점제를 함유할 수 있는, 사용 직전에 멸균 주사가능한 용액 또는 분산액으로 재구성될 수 있는, 하나 이상의 제약상 허용되는 멸균 등장성 수성 또는 비수성 용액, 분산액, 현탁액 또는 에멀젼, 또는 멸균 분말과 조합으로 포함할 수 있다. 본 발명의 제약 조성물에 채용될 수 있는 적합한 수성 및 비수성 담체의 예로는 물, 에탄올, 폴리올 (예컨대 글리세롤, 프로필렌 글리콜, 폴리에틸렌 글리콜 등), 및 이들의 적합한 혼합물, 식물성 오일, 예컨대 올리브유, 및 주사가능한 유기 에스테르, 예컨대 에틸 올레에이트를 들 수 있다. 예를 들어, 코팅 물질, 예컨대 레시틴의 사용에 의해, 분산액의 경우 필요한 입자 크기의 유지에 의해, 및 계면활성제의 사용에 의해 적절한 유동성이 유지될 수 있다.
이들 조성물은 또한 아쥬반트, 예컨대 보존제, 습윤제, 유화제 및 분산제를 함유할 수 있다. 대상 올리고머에 대한 미생물의 작용의 방지는 다양한 항균제 및 항진균제, 예를 들어 파라벤, 클로로부탄올, 페놀 소르브산 등의 도입에 의해 보증될 수 있다. 또한, 등장화제, 예컨대 당, 염화나트륨 등이 조성물 내에 포함되는 것이 바람직할 수 있다. 또한, 주사가능한 제약 형태의 연장된 흡수는 알루미늄 모노스테아레이트 및 젤라틴과 같은 흡수를 지연시키는 제제의 도입에 의해 일어날 수 있다.
일부의 경우, 약물의 효과를 연장하기 위해, 피하 또는 근육내 주사로부터 약물의 흡수를 감속시키는 것이 바람직하다. 이는 관련 기술분야에서 공지된 다른 방법 중에서, 빈약한 수 용해성을 갖는 결정질 또는 비정질 물질의 액체 현탁액의 사용을 수반할 수 있다. 그러면, 약물의 흡수 속도는 그의 용해 속도에 의존하며, 결국 결정 크기 및 결정질 형태에 의존할 수 있다. 대안적으로, 비경구-투여되는 약물 형태의 지연된 흡수는 약물을 오일 비히클에 용해 또는 현탁시킴으로써 달성된다.
주사가능한 데포 형태는 생체분해성 중합체, 예컨대 폴리락티드-폴리글리콜리드에서 대상 올리고머의 미소캡슐 매트릭스를 형성함으로써 제조될 수 있다. 올리고머 대 중합체의 비율, 및 채용되는 특정 중합체의 성질에 따라, 올리고머 방출의 속도는 제어될 수 있다. 다른 생체분해성 중합체의 예로는 폴리(오르토에스테르) 및 폴리(무수물)을 들 수 있다. 데포 주사가능한 제제는 또한 약물을 신체 조직과 혼화성인 리포솜 또는 마이크로에멀젼에서 포획함으로써 제조될 수 있다.
본 발명의 올리고머가 약제로서 인간 및 동물에게 투여될 경우, 이는 그 자체로 또는 예를 들어 0.1 내지 99% (보다 바람직하게는, 10 내지 30%)의 활성 성분을 제약상 허용되는 담체와 조합으로 함유하는 제약 조성물로서 주어질 수 있다.
상기 주목된 바와 같이, 본 발명의 제제 또는 조제물은 경구적으로, 비경구적으로, 전신적으로, 국소적으로, 직장으로 또는 근육내 투여로 주어질 수 있다. 이는 전형적으로 각각의 투여 경로에 적합한 형태로 주어진다. 예를 들어, 이는 정제 또는 캡슐 형태로 주사, 주입 또는 흡입에 의한 주사, 흡입, 안약, 연고, 좌제 등의 투여에 의해; 로션 또는 연고에 의한 국소; 및 좌제에 의한 직장으로 투여된다.
본원에 사용된 어구 "비경구 투여" 및 "비경구적으로 투여되는"은 통상적으로 주사에 의한, 장관 및 국소 투여 이외의 투여 방식을 의마하며, 제한 없이 정맥내, 근육내, 동맥내, 경막내, 관절낭내, 안와내, 심장내, 진피내, 복강내, 경기관내, 피하, 표피하, 관절내, 피막하, 지주막하, 척주내 및 흉골내 주사 및 주입을 들 수 있다.
본원에 사용된 어구 "전신 투여", "전신적으로 투여되는", "말초 투여" 및 "말초적으로 투여되는"은 화합물, 약물 또는 다른 물질의, 그것이 환자의 계로 들어가고, 따라서 물질대사 및 다른 프로세스를 겪도록 하는 중추 신경계 내로 직접적 이외의 투여, 예를 들어 피하 투여를 의미한다.
선택되는 투여 경로와 무관하게, 적합한 수화된 형태로 사용될 수 있는 본 발명의 올리고머, 및/또는 본 발명의 제약 조성물은 관련 기술분야의 통상의 기술자에게 공지된 통상적인 방법에 의해 제약상 허용되는 투여 형태로 제제화될 수 있다. 본 발명의 제약 조성물에서의 활성 성분의 실제 투여 수준은 환자에 대한 수용할 수 없는 독성 없이, 특정 환자, 조성물, 및 투여 방식에 대한 바람직한 치료 반응을 달성하는 데 효과적인 활성 성분의 양을 달성하도록 다양할 수 있다.
선택되는 투여 수준은 채용되는 본 발명의 특정 올리고머 또는 그의 에스테르, 염 또는 아미드의 활성, 투여 경로, 투여 시간, 채용되는 특정 올리고머의 배설 또는 물질대사, 흡수의 속도 및 정도, 치료의 기간, 채용되는 특정 올리고머와 조합으로 사용되는 다른 약물, 화합물 및/또는 물질, 치료되는 환자의 연령, 성별, 체중, 상태, 일반적 건강 및 이전의 병력을 포함하는 다양한 인자, 및 의학 기술분야에서 널리 공지된 유사 인자에 의존할 것이다.
관련 기술분야에서 통상의 기술을 갖는 의사 또는 수의사는 필요한 제약 조성물의 유효량을 용이하게 결정 및 처방할 수 있다. 예를 들어, 의사 또는 수의사는 바람직한 치료 효과를 달성하기 위해 필요한 것보다 낮은 수준으로 제약 조성물에서 채용되는 본 발명의 화합물의 용량을 개시하고, 바람직한 효과를 달성할 때까지 투여량을 점차 증가시킬 수 있다. 일반적으로, 본 발명의 화합물의 적합한 일일 용량은 치료 효과를 생성하는 데 효과적인 최저 용량인 화합물의 양일 것이다. 이러한 유효 용량은 일반적으로 상기 기재된 인자에 의존할 것이다. 일반적으로, 지시된 효과를 위해 사용될 경우 환자에 대한 본 발명의 화합물의 경구, 정맥내, 뇌실내, 근육내 및 피하 용량은 1일 당 체중 킬로그램 당 약 0.0001 내지 약 100 mg의 범위일 것이다.
본 발명의 올리고머 (예를 들어, 포스포로디아미데이트 모르폴리노 올리고머; 에테플러센)의 바람직한 용량은 일반적으로 약 20-100 mg/kg로 투여된다. 일부 경우에, 100 mg/kg 초과의 용량이 필요할 수 있다. 정맥내 투여의 경우, 바람직한 용량은 약 0.5 mg 내지 100 mg/kg이다. 일부 실시양태에서, 올리고머는 약 20 mg/kg, 21 mg/kg, 25 mg/kg, 26 mg/kg, 27 mg/kg, 28 mg/kg, 29 mg/kg, 30 mg/kg, 31 mg/kg, 32 mg/kg, 33 mg/kg, 34 mg/kg, 35 mg/kg, 36 mg/kg, 37 mg/kg, 38 mg/kg, 39 mg/kg, 40 mg/kg, 41 mg/kg, 42 mg/kg, 43 mg/kg, 44 mg/kg, 45 mg/kg, 46 mg/kg, 47 mg/kg, 48 mg/kg, 49 mg/kg 50 mg/kg, 51 mg/kg, 52 mg/kg, 53 mg/kg, 54 mg/kg, 55 mg/kg, 56 mg/kg, 57 mg/kg, 58 mg/kg, 59 mg/kg, 60 mg/kg, 65 mg/kg, 70 mg/kg, 75 mg/kg, 80 mg/kg, 85 mg/kg, 90 mg/kg, 95 mg/kg, 100 mg/kg (이들 사이의 모든 정수 포함)의 용량으로 투여된다. 바람직한 실시양태에서, 올리고머는 30 mg/kg으로 투여된다. 또 다른 바람직한 실시양태에서, 올리고머는 50 mg/kg으로 투여된다.
바람직할 경우, 활성 화합물의 유효 1일 용량은 임의로 단위 투여 형태에서 하루를 통해 적절한 간격으로 개별적으로 투여되는 2, 3, 4, 5, 6 이상의 하위-용량으로서 투여될 수 있다. 특정 상황에서, 용량은 1일 당 한 번의 투여이다. 특정 실시양태에서, 용량은 필요에 따라 기능성 디스트로핀 단백질의 바람직한 발현을 유지하기 위한 매 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14일, 또는 매 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12주, 또는 매 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12개월 당 하나 이상의 투여이다.
일부 실시양태에서, 본 발명의 올리고머 (예를 들어, 포스포로디아미데이트 모르폴리노 올리고머; 에테플러센)는, 일반적으로 규칙적인 간격으로 (예를 들어, 매일, 매주, 격주로, 매달, 격달로) 투여된다. 올리고머는 규칙적인 간격으로, 예를 들어, 매일; 2일에 1회; 3일에 1회; 3일 내지 7일에 1회; 3일 내지 10일에 1회; 7일 내지 10일에 1회; 주 1회; 2주마다 1회; 매달 1회 투여될 수 있다. 예를 들어, 올리고머는 매주 1회 정맥내 주입으로 투여될 수 있다. 올리고머는 보다 장기간에 걸쳐, 예를 들어, 수 주, 수 개월 또는 수 년동안 간헐적으로 투여될 수 있다. 예를 들어, 올리고머는 매 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11 또는 12개월 당 1회 투여될 수 있다. 또한, 올리고머는 매 1, 2, 3, 4 또는 5년 당 1회 투여될 수 있다. 항생제, 스테로이드 또는 다른 치료제의 투여가 상기 투여와 함께 이루어질 수 있거나 또는 후속될 수 있다. 치료 요법은 치료 중인 대상체에 대한 면역검정, 다른 생화학적 시험 및 생리학적 검사의 결과에 기초하여 지시된 바에 따라 조정될 수 있다(용량, 빈도, 경로, 등).
핵산 분자는 본원에 기재되고 관련 기술분야에서 공지된 바와 같이, 전리요법에 의한, 또는 다른 비히클, 예컨대 히드로겔, 시클로덱스트린, 생체분해성 나노캡슐 및 생체결합성 미소구 내로의 혼입에 의한 리포솜 내의 캡슐화를 포함하나 이에 제한되지 않는, 관련 기술분야의 숙련인에게 공지된 다양한 방법에 의해 세포에 투여될 수 있다. 특정 실시양태에서, 마이크로에멀젼화 기술은 친지질성 (수불용성) 제약 제제의 생체분해성을 개선하기 위해 이용될 수 있다. 그 예로는 트리메트린 (Dordunoo, S. K., et al., Drug Development and Industrial Pharmacy, 17(12), 1685-1713, 1991) 및 REV 5901 (Sheen, P. C., et al., J Pharm Sci 80(7), 712-714, 1991)을 들 수 있다. 다른 이점 중에서, 마이크로에멀젼화는 순환계 대신 림프계에 대한 흡수를 우선적으로 지정함으로써 간을 우회하고, 간담즙성 순환에서 화합물의 파괴를 방지함으로써, 증진된 생체이용률을 제공한다.
본 발명의 한 측면에서, 제제는 본원에 제공된 바와 같은 올리고머 및 적어도 하나의 양친매성 담체로부터 형성된 미셀을 함유하며, 여기서 미셀은 약 100 nm 미만의 평균 직경을 갖는다. 보다 바람직한 실시양태는 약 50 nm 미만의 평균 직경을 갖는 미셀을 제공하며, 보다 더 바람직한 실시양태는 약 30 nm 미만, 또는 심지어 약 20 nm 미만의 평균 직경을 갖는 미셀을 제공한다.
모든 적합한 양친매성 담체가 고려되는 반면, 본원에서 바람직한 담체는 일반적으로, 일반적으로 안전한 것으로서 인지된 (Generally-Recognized-as-Safe (GRAS)) 상태를 가지며, 본 발명의 화합물을 가용화하고, 용액이 복합 수상 (예컨대 인간 위장관에서 발견되는 것)과 접촉할 경우 후속 단계에서 이를 마이크로에멀젼화할 수 있는 것이다. 통상적으로, 이들 요건을 만족시키는 양친매성 성분은 2 내지 20의 HLB (친수성 대 친지질성 밸런스) 값을 가지며, 이들의 구조는 C-6 내지 C-20의 범위의 직쇄 지방족 라디칼을 함유한다. 그 예는 폴리에틸렌-글리콜화된 지방 글리세리드 및 폴리에틸렌 글리콜이다.
양친매성 담체의 예로는 포화 및 모노불포화 폴리에틸렌글리콜화된 지방산 글리세리드, 예컨대 완전히 또는 부분적으로 수소화된 다양한 식물성 오일로부터 얻어진 것을 들 수 있다. 이러한 오일은 유리하게는 카르프산 4 내지 10, 카프르산 3 내지 9, 라우르산 40 내지 50, 미리스트산 14 내지 24, 팔미트산 4 내지 14 및 스테아르산 5 내지 15%를 포함하는 특히 바람직한 지방산 조성을 갖는, 트리-, 디- 및 모노-지방산 글리세리드 및 상응하는 지방산의 디- 및 모노-폴리에틸렌글리콜 에스테르로 이루어질 수 있다. 또 다른 유용한 부류의 양친매성 담체로는 포화 또는 모노-불포화 지방산 (스팬 (SPAN)-계열) 또는 상응하는 에톡실화 유사체 (트윈 (TWEEN)-계열)를 갖는, 부분적으로 에스테르화된 소르비탄 및/또는 소르비톨을 들 수 있다.
겔루시르 (Gelucire)-계열, 라브라필 (Labrafil), 라브라솔 (Labrasol) 또는 라우로글리콜 (Lauroglycol) (모두 프랑스 생 프리에스트 소재 가테포세 코포레이션 (Gattefosse Corporation)에 의해 제조 및 유통됨), PEG-모노-올레에이트, PEG-디-올레에이트, PEG-모노-라우레이트 및 디-라우레이트, 레시틴 (Lecithin), 폴리소르베이트 (Polysorbate) 80 등 (미국 및 전세계의 다수의 회사에 의해 생산 및 유통됨)을 포함하는, 시판되는 양친매성 담체가 특히 유용할 수 있다.
특정 실시양태에서, 전달은 본 발명의 조성물을 적합한 숙주 세포 내로 도입하기 위한 리포솜, 나노캡슐, 미소입자, 미소구, 액체 입자, 베지클 등의 사용에 의해 일어날 수 있다. 특히, 본 발명의 조성물은 액체 입자, 리포솜, 베지클, 나노구, 나노입자 등에 캡슐화되어 전달을 위해 제제화될 수 있다. 제제 및 이러한 전달 비히클의 사용은 공지된 및 통상적인 기술을 사용하여 수행될 수 있다.
본 발명에 사용하기에 적합한 친수성 중합체는 용이하게 수용성이고, 베지클-형성 지질에 공유적으로 부착될 수 있고, 독성 효과 없이 생체내에서 내성화되는 (즉, 생체적합성인) 것이다. 적합한 중합체로는 폴리에틸렌 글리콜 (PEG), 폴리락트산 (또한 폴리락티드로도 지칭됨), 폴리글리콜산 (또한 폴리글리콜리드로도 지칭됨), 폴리락트산-폴리글리콜산 공중합체 및 폴리비닐 알코올을 들 수 있다. 특정 실시양태에서, 중합체는 약 100 또는 120 달톤 내지 약 5,000 또는 10,000 달톤, 또는 약 300 달톤 내지 약 5,000 달톤의 분자량을 갖는다. 다른 실시양태에서, 중합체는 약 100 내지 5,000 달톤의 분자량을 갖는, 또는 약 300 내지 5,000 달톤의 분자량을 갖는 폴리에틸렌글리콜이다. 특정 실시양태에서, 중합체는 750 달톤의 폴리에틸렌글리콜 (PEG(750))이다. 중합체는 또한 그 안의 단량체의 수에 의해 정의될 수 있으며, 본 발명의 바람직한 실시양태는 적어도 약 3개의 단량체의 중합체, 예컨대 3개의 단량체로 이루어진 PEG 중합체 (대략 150 달톤)를 이용한다.
본 발명에 사용하기에 적합할 수 있는 다른 친수성 중합체로는 폴리비닐피롤리돈, 폴리메톡사졸린, 폴리에틸옥사졸린, 폴리히드록시프로필 메타크릴아미드, 폴리메타크릴아미드, 폴리디메틸아크릴아미드, 및 유도체화된 셀룰로스, 예컨대 히드록시메틸셀룰로스 또는 히드록시에틸셀룰로스를 들 수 있다.
특정 실시양태에서, 본 발명의 제제는 폴리아미드, 폴리카르보네이트, 폴리알킬렌, 아크릴산 및 메타크릴산 에스테르의 중합체, 폴리비닐 중합체, 폴리글리콜리드, 폴리실록산, 폴리우레탄 및 그의 공중합체, 셀룰로스, 폴리프로필렌, 폴리에틸렌, 폴리스티렌, 락트산 및 글리콜산의 중합체, 폴리무수물, 폴리(오르토)에스테르, 폴리(부트산), 폴리(발레르산), 폴리(락티드-코-카프로락톤), 다당류, 단백질, 폴리히알루론산, 폴리시아노아크릴레이트, 및 이들의 블렌드, 혼합물 또는 공중합체로 이루어진 군으로부터 선택되는 생체적합성 중합체를 포함한다.
시클로덱스트린은 각각 그리스 문자 α, β 또는 γ에 의해 지정되는 6, 7 또는 8개의 글루코스 단위로 이루어진 시클릭 올리고당류이다. 글루코스 단위는 α-1,4-글루코시드 결합에 의해 연결된다. 당 단위의 쇄 형태의 결과로서, 모든 2차 히드록실기 (C-2, C-3에서)는 고리의 한 측면에 위치되는 반면, C-6에서의 모든 1차 히드록실기는 다른 측면에 위치된다. 결과로서, 외부 면은 시클로덱스트린을 수용성으로 만드는 친수성이다. 반대로, 시클로덱스트린의 공동은, 이들이 원자 C-3 및 C-5의 수소에 의해 및 에테르-유사 산소에 의해 라이닝되기 때문에 소수성이다. 이들 매트릭스는 예를 들어 스테로이드 화합물, 예컨대 17α-에스트라디올을 포함하는, 다양한 상대적으로 소수성 화합물과 복합체화를 허용한다 (예를 들어, 문헌 [van Uden et al. Plant Cell Tiss. Org. Cult. 38:1-3-113 (1994)] 참조). 복합체화는 반 데르 발스 상호작용 및 수소 결합 형성에 의해 일어난다. 시클로덱스트린의 화학의 일반적인 검토를 위해서는, 문헌 [Wenz, Agnew. Chem. Int. Ed. Engl., 33:803-822 (1994)]을 참조한다.
시클로덱스트린 유도체의 물리-화학적 특성은 치환의 종류 및 정도에 강하게 의존한다. 예를 들어, 수중에서의 이들의 용해도는 불용성 (예를 들어, 트리아세틸-베타-시클로덱스트린) 내지 147% 가용성 (w/v) (G-2-베타-시클로덱스트린)의 범위이다. 또한, 이들은 많은 유기 용매에 가용성이다. 시클로덱스트린의 특성은 그들의 용해도를 증가 또는 감소시킴으로써 다양한 제제 성분의 용해도에 걸쳐 제어를 가능하게 한다.
다수의 시클로덱스트린 및 그의 제조 방법이 기재되었다. 예를 들어 파메터 (I) 등 (Parmeter (I), et al.) (미국 특허 제3,453,259호) 및 그라메라 등 (Gramera, et al.) (미국 특허 제3,459,731호)은 전기중성적 시클로덱스트린을 기재하였다. 다른 유도체로는 양이온성 특성을 갖는 시클로덱스트린 [파메터 (II), 미국 특허 제3,453,257호], 불용성 가교된 시클로덱스트린 (솔름즈 (Solms), 미국 특허 제3,420,788호), 및 음이온성 특성을 갖는 시클로덱스트린 [파메터 (III), 미국 특허 제3,426,011호]을 들 수 있다. 음이온성 특성을 갖는 시클로덱스트린 유도체 중에서, 카르복실산, 아인산, 포스피너스산, 포스폰산, 인산, 티오포스폰산, 티오술핀산 및 술폰산은 모 시클로덱스트린에 부속될 수 있다 [파메터 (III), 상기 문헌 참조]. 또한, 술포알킬 에테르 시클로덱스트린 유도체는 스텔라 등 (Stella, et al.) (미국 특허 제5,134,127호)에 의해 기재되었다.
리포솜은 수성 내부 구획을 둘러싸는 적어도 하나의 지질 이중층 막으로 이루어진다. 리포솜은 막 유형 및 크기에 의해 특성화될 수 있다. 소형 유니라멜라 베지클 (SUV)은 단일 막을 가지며, 전형적으로 직경 0.02 내지 0.05 ㎛ 범위이고, 대형 유니라멜라 베지클 (LUVS)은 전형적으로 0.05 ㎛ 초과이다. 올리고라멜라 대형 베지클 및 다중라멜라 베지클은 다중의 통상적으로 동심원의 막 층을 가지며, 전형적으로 0.1 ㎛ 초과이다. 몇몇 비동심원 막을 갖는 리포솜, 즉 보다 대형 베지클 내에 함유된 몇몇 보다 소형 베지클은 다중베지클성 베지클로 지칭된다.
본 발명의 한 측면은 본 발명의 올리고머를 함유하는 리포솜을 포함하는 제제에 관한 것이며, 여기서 리포솜 막은 증가된 운반 능력을 갖는 리포솜을 제공하도록 제제화된다. 대안적으로 또는 추가로, 본 발명의 화합물은 리포솜의 리포솜 이중층 내에 함유되거나, 그 위로 흡수될 수 있다. 본 발명의 올리고머는 지질 계면활성제와 응집되고, 리포솜의 내부 공간 내로 운반될 수 있으며, 이들의 경우, 리포솜 막은 활성제-계면활성제 응집체의 파괴 효과를 저항하도록 제제화된다.
본 발명의 한 실시양태에 따라, 리포솜의 지질 이중층은 폴리에틸렌 글리콜 (PEG)로 유도체화된 지질을 함유하여, PEG 쇄는 지질 이중층의 내표면으로부터 리포솜에 의해 캡슐화된 내부 공간으로 연장되며, 지질 이중층의 외부로부터 주변 환경으로 연장된다.
본 발명의 리포솜 내에 함유된 활성제는 가용화된 형태이다. 계면활성제 및 활성제의 응집체 (예컨대 관심의 활성제를 함유하는 에멀젼 또는 미셀)는 본 발명에 따른 리포솜의 내부 공간 내에 포획될 수 있다. 계면활성제는 활성제를 분산 및 가용화시키는 작용을 하며, 다양한 쇄 길이 (예를 들어, 약 C14 내지 약 C20)의 생체적합성 리소포스파티딜콜린 (LPG)을 포함하나 이에 제한되지 않는 임의의 적합한 지방족, 지환족 또는 방향족 계면활성제로부터 선택될 수 있다. 중합체-유도체화된 지질, 예컨대 PEG-지질은 또한 이들이 미셀/막 융합을 억제하도록 작용하고, 중합체의 계면활성제 분자로의 첨가가 미셀 형성에서 계면활성제 및 보조제의 CMC를 감소시키는 것과 같이 미셀 형성에 이용될 수 있다. 마이크로몰 범위의 CMO를 갖는 계면활성제가 바람직하며, 보다 높은 CMC 계면활성제는 본 발명의 리포솜 내에서 포획되는 미셀을 제조하는 데 이용될 수 있다.
본 발명에 따른 리포솜은 관련 기술분야에서 공지된 임의의 다양한 기술에 의해 제조될 수 있다. 예를 들어, 미국 특허 제4,235,871호; 공개된 PCT 출원 WO 96/14057; 문헌 [New RRC, Liposomes: A practical approach, IRL Press, Oxford (1990), pages 33-104]; [Lasic DD, Liposomes from physics to applications, Elsevier Science Publishers BV, Amsterdam, 1993]을 참조한다. 예를 들어, 본 발명의 리포솜은 친수성 중합체로 유도체화된 지질을 예비형성된 리포솜 내로 확산시킴으로써, 예컨대 리포솜에서 바람직한 유도체화된 지질의 최종 몰 %에 상응하는 지질 농도로, 예비형성된 리포솜을 지질-그래프팅된 중합체로 이루어진 미셀에 노출시킴으로써 제조될 수 있다. 친수성 중합체를 함유하는 리포솜은 또한 관련 기술분야에서 공지된 바와 같은 균질화, 지질-장 수화 또는 압출 기술에 의해 형성될 수 있다.
또 다른 예시적인 제제화 과정에서, 활성제는 먼저 소수성 분자를 용이하게 가용화하는 리소포스파티딜콜린 또는 다른 저 CMC 계면활성제 (중합체 그래프팅된 지질을 포함함)에서의 초음파처리에 의해 분산된다. 생성된 활성제의 미셀 현탁액은 그 후 적합한 몰 %의 중합체-그래프팅된 지질, 또는 콜레스테롤을 함유하는 건조된 지질 샘플을 재수화하는 데 사용된다. 지질 및 활성제 현탁액은 그 후 관련 기술분야에서 공지된 압출 기술을 이용하여 리포솜으로 형성되며, 생성된 리포솜은 표준 칼럼 분리에 의해 비캡슐화된 용액으로부터 분리된다.
본 발명의 한 측면에서, 리포솜은 선택된 크기 범위에서 실질적으로 균질한 크기를 갖도록 제조된다. 하나의 효과적인 사이징 방법은 선택된 균일한 공극 크기를 갖는 일련의 폴리카르보네이트 막을 통해 리포솜의 수성 현탁액을 압출하는 것을 포함하며, 막의 공극 크기는 그 막을 통한 압출에 의해 제조되는 리포솜의 가장 큰 크기와 대략 상응할 것이다. 예를 들어, 미국 특허 제4,737,323호 (1988년 4월 12일)를 참조한다. 특정 실시양태에서, 다르마FECT (DharmaFECT)® 및 리포펙타민 (Lipofectamine)®과 같은 시약이 폴리뉴클레오티드 또는 단백질을 세포 내로 도입하는 데 이용될 수 있다.
본 발명의 제제의 방출 특성은 캡슐화 물질, 캡슐화된 약물의 농도, 및 방출 변형제의 존재에 의존한다. 예를 들어, 방출은 예를 들어 위에서와 같이 낮은 pH에서, 또는 장에서와 같이 보다 높은 pH에서만 방출하는 pH 민감성 코팅을 이용하여 pH 의존성이 되도록 조작될 수 있다. 장용성 코팅은 위를 통한 통과 후까지 방출의 발생을 방지하는 데 사용될 수 있다. 상이한 물질 내에 캡슐화된 시안아미드의 다중 코팅 또는 혼합물은 위에서 초기 방출을 얻은 후, 장에서 후기 방출을 얻는 데 사용될 수 있다. 방출은 또한 캡슐로부터의 확산에 의해 약물의 물 흡수 또는 방출을 증가시킬 수 있는 염 또는 공극 형성제의 포함에 의해 조작될 수 있다. 약물의 용해도를 변형하는 부형제는 또한 방출 속도를 제어하는 데 사용될 수 있다. 매트릭스의 분해 또는 매트릭스로부터의 방출을 증진시키는 제제가 또한 혼입될 수 있다. 이는 개별 상으로서 (즉, 입자로서) 첨가되는 약물에 첨가될 수 있거나, 화합물에 의존하는 중합체 상에 공동-용해될 수 있다. 대개의 경우, 양은 0.1% 내지 30% (w/w 중합체)일 것이다. 분해 증진제의 유형으로는 무기염, 예컨대 황산암모늄 및 염화암모늄, 유기산, 예컨대 시트르산, 벤조산 및 아스코르브산, 무기 염기, 예컨대 탄산나트륨, 탄산칼륨, 탄산칼슘, 탄산아연 및 수산화아연, 및 유기 염기, 예컨대 프로타민 술페이트, 스페르민, 콜린, 에탄올아민, 디에탄올아민 및 트리에탄올아민 및 계면활성제, 예컨대 트윈 (Tween)® 및 플루로닉 (Pluronic)®을 들 수 있다. 매트릭스에 미소구조를 첨가하는 공극 형성제 (즉, 수용성 화합물, 예컨대 무기염 및 당)은 입자로서 첨가된다. 범위는 전형적으로 1% 내지 30% (w/w 중합체)이다.
흡수는 또한 장 내의 입자의 체류 시간을 변경함으로써 조작될 수 있다. 이는 예를 들어 점막 접착성 중합체로 입자를 코팅함으로써, 또는 캡슐화 물질로서 선택함으로써 달성될 수 있다. 그 예로는 유리 카르복실기를 갖는 대부분의 중합체, 예컨대 키토산, 셀룰로스, 특히 폴리아크릴레이트 (본원에 사용된 폴리아크릴레이트는 아크릴레이트기 및 변형된 아크릴레이트기, 예컨대 시아노아크릴레이트 및 메타크릴레이트를 포함하는 중합체를 지칭함)를 들 수 있다.
올리고머는 외과적 또는 의학적 장치 또는 이식물에 의해 그 내에 함유되거나 방출에 적합화되도록 제제화될 수 있다. 특정 측면에서, 이식물은 올리고머로 코팅되거나, 다르게는 처리될 수 있다. 예를 들어, 히드로겔, 또는 다른 중합체, 예컨대 생체적합성 및/또는 생체분해성 중합체는 본 발명의 조성물로 이식물을 코팅하는 데 사용될 수 있다 (즉, 조성물은 히드로겔 또는 다른 중합체를 사용함으로써 의학적 장치로 사용하는 데 적합화될 수 있음). 제제로 의학적 장치를 코팅하기 위한 중합체 및 공중합체는 관련 기술분야에서 널리 공지되어 있다. 이식물의 예로는 스텐트, 약물-용리 스텐트, 봉합물, 보철, 혈관 카테터, 투석 카테터, 혈관 그래프트, 인공 심장 판막, 심장 박동 조율기, 삽입형 제세동기, IV 바늘, 골 세팅 및 형성을 위한 장치, 예컨대 핀, 스크루, 플레이트, 및 다른 장치, 및 상처 치유를 위한 인공 조직 매트릭스를 들 수 있으나, 이에 제한되지는 않는다.
본원에 제공된 방법 외에, 본 발명에 따라 사용하기 위한 올리고머는 다른 약제와의 유사성에 의해, 인간 또는 동물 의약에 사용하기 위해 임의의 편리한 방법으로 투여용으로 제제화될 수 있다. 안티센스 올리고머 및 그의 상응하는 제제는 단독으로 또는 근육 이영양증의 치료에서의 다른 치료 전략, 예컨대 근모세포 이식, 줄기 세포 요법, 아미노글리코시드 항생제의 투여, 프로테아좀 억제제, 및 상향-조절 요법 (예를 들어, 유트로핀의 상향조절, 디스트로핀의 상염색체 파라로그)과 조합으로 투여될 수 있다.
숙련된 의사는 임의의 특정한 동물 및 상태를 위한 최적의 투여 경로 및 임의의 투여량을 용이하게 결정할 수 있을 것이기 때문에, 기재된 투여 경로는 안내로서만 의도된다. 기능성 신규 유전 물질을 세포 내로 시험관내 및 생체내 둘 다로 도입하는 다수의 접근이 시도되었다 (Friedmann (1989) Science, 244:1275-1280). 이들 접근으로는 변형된 레트로바이러스 내로 발현되는 유전자의 통합 (Friedmann (1989) 상기 문헌; Rosenberg (1991) Cancer Research 51(18), suppl.: 5074S-5079S); 비-레트로바이러스 벡터 (예를 들어, 아데노-연관 바이러스 벡터) 내로의 통합 (Rosenfeld, et al. (1992) Cell, 68:143-155; Rosenfeld, et al. (1991) Science, 252:431-434); 또는 리포솜을 통한 이종 프로모터-인핸서 요소에 연결된 트랜스진의 전달 (Friedmann (1989), 상기 문헌; Brigham, et al. (1989) Am. J. Med. Sci., 298:278-281; Nabel, et al. (1990) Science, 249:1285-1288; Hazinski, et al. (1991) Am. J. Resp. Cell Molec. Biol., 4:206-209; 및 Wang and Huang (1987) Proc. Natl. Acad. Sci. (USA), 84:7851-7855); 리간드-특이적, 양이온-기재 수송 시스템으로의 커플링 (Wu and Wu (1988) J. Biol. Chem., 263:14621-14624) 또는 네이키드 DNA, 발현 벡터의 사용 (Nabel et al. (1990), 상기 문헌); Wolff et al. (1990) Science, 247:1465-1468)을 들 수 있다. 트랜스진의 조직 내로의 직접 주입은 국재화된 발현만을 생성한다 (Rosenfeld (1992) 상기 문헌); Rosenfeld et al. (1991) 상기 문헌; Brigham et al. (1989) 상기 문헌; Nabel (1990) 상기 문헌; 및 Hazinski et al. (1991) 상기 문헌). 브리그함 등 (Brigham et al.) 그룹 (Am. J. Med. Sci. (1989) 298:278-281 및 Clinical Research (1991) 39 (abstract))은 DNA 리포솜 복합체의 정맥내 또는 동맥내 투여 후의 마우스의 폐만의 생체내 형질감염을 보고하였다. 인간 유전자 요법 과정의 검토 문헌의 예는 문헌 [Anderson, Science (1992) 256:808-813]이다.
V. 키트
본 발명은 또한 그의 사용을 위한 지시서와 함께 적합한 용기에 패키징된 적어도 안티센스 분자 (예를 들어, 본원의 표 3 및 4에 개시된 바와 같은 디스트로핀 유전자의 엑손 1-79 중 임의의 하나 이상; 예를 들어, 엑손 51에 특이적으로 혼성화할 수 있는 하나 이상의 안티센스 올리고뉴클레오티드)를 포함하는, 유전적 질환을 갖는 환자의 치료를 위한 키트를 제공한다. 키트는 또한 보조 시약, 예컨대 완충액, 안정화제 등을 함유할 수 있다. 관련 기술분야의 통상의 기술자는 상기 방법의 적용이 많은 다른 질환의 치료에 사용하기에 적합한 안티센스 분자를 확인하기 위한 폭넓은 적용을 가짐을 이해해야 한다.
VII.
실시예
물질 및 방법
환자
적격한 환자는 7세에서 13세 사이 (7세 및 13세 포함)로서, 엑손 51의 스키핑에 의해 교정될 수 있었던 DMD 유전자의 아웃-오브-프레임 결실을 가진 환자였다. 환자들은 안정한 심장 및 폐 기능을 가졌고 등록 전에 적어도 24주 동안 안정한 용량의 글루코코르티코이드 투여를 받은 것으로 확인되었다. 기준선에서 6-분 보행 시험 (6MWT)시 200 내지 400 미터 (±10%)를 걸을 수 있는 환자만이 등록되었다.
연구 설계
이 1년 시험은 2 상으로 수행되었다: (1) 치료는 제24주까지 이중-맹검이었고, (2) 그 후에 개방-표지였다. 1차 종점은 디스트로핀 섬유 비율의 변화 및 6-분 보행 시험 (6MWT)으로 측정된 보행이었다.
연구 201은 에테플러센에 대한 단일-부위, 무작위, 이중-맹검, 위약-대조, 다중-용량의 효능, 안전성 및 내약성 시험이었다. 12명의 DMD 환자를 하기 3개 군 중 하나로 무작위 배정하였다: 에테플러센 30 mg/kg/주 (코호트 1); 에테플러센 50 mg/kg/주 (코호트 2); 또는 위약/지연 에테플러센 (코호트 3). 모든 환자는 매주 정맥내 에테플러센 또는 위약/지연 에테플러센 투여를 받았다. 위약-치료 환자는 제25주에 매주 에테플러센 30 (n=2) 또는 50 mg/kg (n=2)로 변경되었다. 효능 및 안전성은 예정된 방문시에 평가되었고, 독립적인 데이터 안전성 모니터링 위원회가 모든 환자의 후생을 보장하였다. 모든 환자는 기준선에서 이두근 생검을 받았다. 50 mg/kg 군 및 2명의 위약-치료 환자에 대해 제12주에 및 30 mg/kg 군 및 2명의 위약-치료 환자에 대해 제24주에 반대쪽 팔 (이두근)에서 후속 생검을 실시하였다.
환자들은 장기, 개방-표지 연장 연구인 연구 202 하에서 30 또는 50 mg/kg 에테플러센으로써 계속해서 매주 투여되었다. 제48주에 모든 환자에서의 세번째 생검 (좌측 삼각근)을 포함하여 모든 효능 평가를 연구 202 동안 계속 실시하였다. 연구 기간 내내 유해 사례에 대한 모니터링을 지속하였다. 연구 설계에 대한 개략도는 도 2에 제시되어 있다.
연구 약물
에테플러센 [서열 5'-CTCCAACATCAAGGAAGATGGCATTTCTAG-3'] (서열 1000)은 사렙타 쎄러퓨틱스, 인코퍼레이티드 (Sarepta Therapeutics, Inc.)에 의해, 일회용 포스페이트-완충 염수 바이알 (100 mg/ml) 중에 공급되었다. 에테플러센을 150 ml 생리 염수로 재구성하고 60분에 걸쳐 주입시켰다. 연구 201의 처음 24주 동안 투여된 위약은 동일한 포스페이트-완충 염수 바이알로서 공급되었고 에테플러센과 동일한 방식으로 투여되었다.
안전성 및
내약성
모니터링
유해 사례, 활력 징후, 신체 검사, 심전도, 심장초음파, 및 임상 실험실 시험에 대한 평가에 의해 안전성을 평가하였다. 또한, 혈청 시스타틴 C 및 소변 시스타틴 C 및 KIM-1에 대한 정기적인 평가를 통해 신장 기능을 모니터링하였다.
약동학 및 면역 평가
에테플러센의 약동학적 파라미터는 인증된 정밀 음이온 교환 고성능 액체 크로마토그래피와 함께 형광 검출 생체분석 방법을 이용하여 제12회 용량 후에 채취된 혈장 및 소변으로부터 확립하였다. 혈장 농도의 분석을 위한 단일 샘플은 제24, 25, 및 36주에 채취하였다. 신규 디스트로핀 단백질에 대한 면역 반응은 이전에 공개된 방법에 따라 ELISPOT을 이용하여 제24주까지 매 6주마다 측정하였다.
생화학적 효능 평가
치료전 및 치료후 디스트로핀 발현 연구는 에테플러센 및 다른 엑손 스키핑 후보들에 대한 이전의 연구에서 사용된 디스트로핀에 대한 고도로 민감한 마커인 MANDYS106 [MDA 모노클로날 항체 라이브러리의 글렌 모리스(Glen Morris)로부터의 기증품]에 기초하였다. 적어도 200 μm 정도 분리된 3개의 10 μm 동결 절편을 MANDYS106으로 염색한 후, 2차 항체 (알렉사 플루오르(Alexa Fluor) 594 염소 항-마우스 항체)로 염색하였다. 양성 섬유의 수를 카운팅된 총 섬유로 나누어 디스트로핀-양성 섬유 비율을 계산하였다. 정상 근육 샘플이 100% 디스트로핀-양성 섬유를 가지므로, 디스트로핀-양성 섬유의 비율은 정상에 대한 백분율로서 표현된다. 바이오퀀드 영상 분석 소프트웨어를 이용한 디스트로핀 정량화를 위해 동일한 항체로 염색된 절편을 사용하였다. 총 디스트로핀 형광 신호 강도는 정상에 대한 백분율로 기록하였다.
지지 측정에는 사르코글리칸 착물의 성분 (β,γ) 발현, 뉴런 NOS, 및 웨스턴 블롯 (노바카스트라(Novacastra)로부터의 항 디스트로핀 항체 NCL-Dys1 이용)이 포함되었다. 엑손 스키핑에 대한 확인을 위해 이전에 기재한 바와 같은 디스트로핀-특이적 역방향 프라이머를 이용하여 400 ng의 총 RNA에 대해 RT-PCR 분석을 실시하였다.
임상 효능 평가
6MWT는 문헌 [McDonald, et al. (Muscle Nerve, 2010; 42:966-74)] (본원에 참조로 포함됨)에 의해 DMD 환자에 대해 확립된 프로토콜을 이용하여 진행되었다. 탐색적인 기능수행 결과에는 노스 스타(North Star) 보행 평가, 정량적 근육 시험, 9-홀 페그(9-Hole Peg) 시험, 폐 기능 시험 (PFT), 시간별 기능 시험, 및 삶의 질 평가가 포함되었다.
통계적 분석
모든 통계적 분석을 위해 SAS 버전 9.3 (미국 노스캐롤라이나 캐리)을 사용하였다. 고정된 효과로서의 치료, 무작위 효과로서의 치료 내에 포함된 대상, 근육 생검 데이터의 분석을 위한 공변량으로서 기준선 값 및 DMD 진단 이후의 시간을 갖는 혼합 모델을 사용하였다. 고정된 효과로서의 치료, 시간, 및 시간에-따른-치료 상호작용 조건, 무작위 효과로서의 치료 내에 포함된 대상, 및 6MWT 데이터의 분석을 위한 공변량으로서 기준선 값 및 DMD 진단 이후의 시간을 갖는 혼합 모델 반복 측정 (MMRM)을 사용하였다. 치료 의향이 있는(intent-to-treat) 집단에 대해 안전성 및 근육 생검 분석을 실시하였다; 6MWT를 비롯한 보행-관련 결과에 대한 분석에서는 등록 주 내에 질환 진행의 징후 및 6MWT시 상당한 저하를 보여 제24주 이상의 보행 측정을 실시할 수 없었던 코호트 1 내의 2명의 환자를 제외한 변경된 치료 의향 (mITT) 집단이 이용되었다.
실시예
1: 대상체
특징
이 연구에서의 12명의 환자의 기준선 특징이 표 2에 요약되어 있다. 엑손 51 스키핑에 순응가능한 5개의 상이한 유전자형이 이 연구 집단에서 제시되었다. 기준선에서 6-분 보행 시험 (6MWT)시의 평균 거리는 DMD를 가진 소아에 대한 다른 연구에서의 것과 유사하였고, 예상된 바와 같이, 동일 연령의 건강한 소아에서 전형적으로 관찰된 600 미터 이상보다 상당히 낮았다. 표본 추출의 추계적 성질에 기인하여, 30 mg/kg 코호트는 다른 코호트에 비해 연령, 신장 및 체중이 약간 더 컸으며, 기준선에서 보다 낮은 평균 6MWT 거리를 가졌다. 모든 환자는 계획된 연구 약물을 모두 주입받았으며, 모든 평가를 완료하였다.
실시예
2: 안전성
및 유해 사례의 부재
에테플러센은 48주 치료 동안 치료-관련 유해 사례, 심각한 유해 사례, 중단 또는 투여 누락없이 잘 용인되었다. 더욱이, 신체 검사시 또는 활력 징후에서 임상적으로 유의한 변화도 관찰되지 않았다. 심전도, 심장초음파, 및 PFT는 안정한 상태로 유지되었고, 화학적 특성은 혈액, 신장, 응고 또는 간 기능에서 임상적으로 유의한 변화를 나타내지 않았다. 경미한 일시적인 단백뇨는 단일 위약-치료 대상체에서 관찰되었다.
실시예
3: 약동학적
프로파일
제12주에 PK 파라미터에 대한 분석 결과, 흡수가 신속한 것으로 나타났다. 혈장 클리어런스는 30 mg/kg의 경우 평균 339 ± 75.8 mL/hr/kg 및 50 mg/kg의 경우 평균 319 ± 125 mL/hr/kg이었다. 반감기는 30 mg/kg의 경우 평균 3.30 ± 0.341 hr 및 50 mg/kg의 경우 평균 3.17 ± 0.249 hr이었으며, 이 때 신장 클리어런스는 총 전신 클리어런스의 대략 65-70%를 차지하였다.
실시예
4: 효능
제48주에, 에테플러센는 디스트로핀 양성 섬유의 수 및 강도의 왕성한 증가를 일으켰다. 도 3에서 나타낸 바와 같이, 48주 동안 중단없이 30 또는 50 mg/kg 에테플러센을 투여받은 환자는 기준선에 비해 디스트로핀-양성 섬유의 비율에 있어 정상의 47%까지의 평균적 증가를 나타내었다(p ≤0.001). 30 (52%; p ≤0.001) 및 50 (43%; p ≤0.008) mg/kg 코호트를 개별적으로 분석했을 때 증가가 유사했는데, 이는 신규 디스트로핀의 제조에 대한 에테플러센의 효과가 이 범위의 용량 내에서는 용량에 무관하다는 것을 시사한다.
치료 지속기간이 신규 디스트로핀 제조에 대해 미치는 영향을 평가하기 위해 시차를 둔 시점에서 생검을 채취하였다 (도 2 참조). 제12주에, 50 mg/kg 코호트는 신규 디스트로핀의 수준이 검출불가능한 정도였다. 제24주에, 30 mg/kg 코호트는 정상의 23%까지의 디스트로핀-양성 섬유의 백분율의 증가를 나타냈으며 (p ≤0.002), 30 또는 50 mg/kg 에테플러센을 이용한 24주 치료 후 제48주에, 위약/지연 에테플러센 코호트 내의 4명의 환자는 기준선에 비해 정상의 38%까지의 증가를 나타냈다(p ≤0.009). 종합하면, 이들 데이터는 신규 디스트로핀 제조를 균일하게 회복시키는 에테플러센의 능력에 있어 치료 지속기간이 중요한 역할을 한다는 것을 시사한다. 이러한 발견과 일치하는 것으로서, 에테플러센은 또한 모든 3개 치료군에서 제48주에 평균 형광 신호 강도를 유의미하게 증가시켰다 (모든 p-값 ≤0.023).
도 4는 디스트로핀-양성 섬유의 백분율에 대한 에테플러센의 시간-의존적 효과를 도시하며 (패널 4a), 이는 또한 근초에서 β- 및 γ-사르코글리칸 및 nNOSμ의 회복을 동반하였다 (패널 4b). 모든 환자에서 웨스턴 블롯 및 RT-PCR에 의해 디스트로핀 발현 및 엑손 스키핑이 확인되었다. 대표적 환자로부터의 RT-PCR 결과가 패널 4c에 제시되어 있다. 이들 데이터는 환자에서 기능적 디스트로핀의 증가를 확증해 주었다.
실시예
5: 기능적
결과
보행 능력의 점진적 소실은 DMD의 일반적인 특징이며, 대부분의 환자들은 7세 또는 8세 경까지 기능상의 손상을 보이며 10세 내지 14세 경에는 휠체어에 의존하게 된다. 이와 일치되는 것으로서, 본 연구에서 위약/지연 에테플러센 코호트에 배정된 소년들은 제12주 후에 이전의 연구에서 예측된 비율로 보행 능력의 저하를 보였으며, 제48주까지 대략 60 미터의 손실에 이르렀다 (도 5). 이와는 현저히 대조적으로, 에테플러센-치료 환자들은 연구 기간에 걸쳐 안정한 보행 거리를 유지하였는데, 제48주까지 기준선으로부터 약 7 미터의 평균 증가를 나타냈다. 에테플러센-치료 환자 및 위약/지연 에테플러센 코호트 내 환자 간의 차이는 제32주에 처음으로 통계학적으로 유의미해졌다 (39 미터 차이; p ≤0.05). 흥미롭게도, 위약/지연 에테플러센 코호트 내 환자들은 제36주 후, 즉, 제25주에 에테플러센으로 치료를 시작한지 12주 및 24주 후에 안정화되는 것으로 나타났다. 이전에 언급하였듯이, 등록 주 내에서 신속한 질환 진행의 징후 및 6MWT에서의 상당한 저하를 보여 24주 이상에서 보행 측정을 실시할 수 없었던 2명의 소년들은 이 분석에서 제외되었다. 그러나, 두 소년 모두 48주 동안 치료-관련 유해 사례없이 에테플러센 투여가 유지되었고, 각각 PFT 및 9-홀 페그 시험으로 측정한 바 안정한 폐 및 상지 기능을 유지하였다.
주목할 만한 것으로서, 48주 동안 에테플러센을 투여받은, 6MWT (n=6)에 대해 평가가능한 환자는, 위약/지연 코호트와 비교하여 6MWT에서 유의미하게 (p ≤0.001) 개선되었다 (67.3m).
두 코호트 모두 하기 기재한 바와 같이 120주에 걸쳐 안정한 6MWT 결과를 유지하였다. 120주에, 6MWT를 수행할 수 있었던 30 mg/kg 및 50 mg/kg 에테플러센 코호트 내 환자 (변경된 치료 의향이 있는 또는 mITT 집단; n=6)는 보행 능력에 있어 기준선으로부터 13.9 미터, 또는 5% 미만의 약간의 저하를 나타내어 대체적 안정성을 경험하였다. mITT 집단의 경우, 24주의 위약 후 제25주에 치료를 개시한 위약/지연-치료 코호트 (n=4)와 비교하여 64.9 미터의 통계학적으로 유의한 치료 이익 (p ≤0.006)이 관찰되었다. (에테플러센으로의 치료 전) 연구 초기에 상당한 저하를 경험한 후, 위약/지연-치료 코호트도 또한 제36주에서 제120주까지 1.5년 초과 동안 보행 능력에서 안정화를 나타냈으며, 그 기간에 유의미한 수준의 디스트로핀이 마찬가지로 생성되었고, 이 기간에 걸쳐 9.5 미터의 감소가 있었다. 이들 분석은 시험을 이틀 연속 실시하였을 때의 최대 6MWT 점수에 기초하였다.
실시예
6: 면역
반응
제24주를 비롯한 평가된 임의의 시점에서 디스트로핀 펩티드 풀(pool) (전체 단백질에 걸쳐 연장됨)에 대한 인터페론-γ-유도된 반점 형성 콜로니의 수에 있어 에테플러센-치료 환자 및 위약-치료 환자 간에 차이가 없었는데, 이는 에테플러센-치료 환자에서 새로이 발현된 디스트로핀이 T 세포 반응을 유도하지 않았음을 시사한다.
실시예
7:
폐 기능
두 투여 코호트 모두에서 최대 흡기압 및 호기압 (MIP 및 MEP)으로 측정한, 기준선에서 제120주까지 호흡근 기능은 MIP에서 평균 14.6% 증가 및 MEP에서 평균 15.0% 증가를 나타냈다. 예측된 MIP % (체중에 대해 조절된 MIP) 및 예측된 MEP % (연령에 대해 조절된 MEP)는 예측된 MIP %에서 기준선에서의 90.2%에서 제120주에 95.2%로의 평균 증가, 및 예측된 MEP %에서 기준선에서의 79.3%에서 제120주에 79.6%로의 약간의 평균 증가를 나타냈다. 또한, 폐 부피의 척도인 강제 폐활량 (FVC)에서 기준선에서 제120주까지 8.7%의 평균 증가가 있었고, 예측된 FVC % (연령 및 신장에 대해 조절된 FVC)는 제120주까지 평균 90% 초과로 유지되었으며, 기준선에서 101% 및 제120주에서 93%였다.
*********************
본 명세서에서 인용된 모든 공개물 및 특허 출원은, 각각의 개별적 공개물 또는 특허 출원이 구체적으로 및 개별적으로 참조로 포함되는 것으로 표시된 것과 마찬가지로 본원에서 참조로 포함된다.
참고문헌
SEQUENCE LISTING
<110> SAREPTA THERAPEUTICS, INC.
<120> IMPROVED COMPOSITIONS FOR TREATING MUSCULAR DYSTROPHY
<130> AVN-012BPC
<140> PCT/US2014/029610
<141> 2014-03-14
<150> US 61/793,463
<151> 2013-03-15
<160> 891
<170> PatentIn version 3.5
<210> 1
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (eteplirsen H51A(+66+95))
<400> 1
ctccaacatc aaggaagatg gcatttctag 30
<210> 2
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H51A(+66+90))
<400> 2
acaucaagga agauggcauu ucuag 25
<210> 3
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H51A(+61+90))
<400> 3
acaucaagga agauggcauu ucuaguuugg 30
<210> 4
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.exon51.25.001.2)
<400> 4
gagcaggtac ctccaacatc aaggaa 26
<210> 5
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H50D(+07-18))
<400> 5
gggauccagu auacuuacag gcucc 25
<210> 6
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (AVI-4038/5038 )
<400> 6
cttacaggct ccaatagtgg tcagt 25
<210> 7
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A27(+30+56))
<400> 7
cctccggttc tgaaggtgtt cttgtac 27
<210> 8
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+36+60) )
<400> 8
gttgcctccg gttctgaagg tgttc 25
<210> 9
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H45A (-03+19))
<400> 9
caatgccatc ctggagttcc tg 22
<210> 10
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H8A(-06+18))
<400> 10
gauagguggu aucaacaucu guaa 24
<210> 11
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H8A (-03+18))
<400> 11
gauagguggu aucaacaucu g 21
<210> 12
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H8A(-07+18))
<400> 12
gauagguggu aucaacaucu guaag 25
<210> 13
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H8A(-06+14))
<400> 13
ggugguauca acaucuguaa 20
<210> 14
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H8A(-10+10))
<400> 14
guaucaacau cuguaagcac 20
<210> 15
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H7A(+45+67))
<400> 15
ugcauguucc agucguugug ugg 23
<210> 16
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H7A(+02+26))
<400> 16
cacuauucca gucaaauagg ucugg 25
<210> 17
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H7D(+15-10))
<400> 17
auuuaccaac cuucaggauc gagua 25
<210> 18
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H7A(-18+03))
<400> 18
ggccuaaaac acauacacau a 21
<210> 19
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (C6A(-10+10))
<400> 19
cauuuuugac cuacaugugg 20
<210> 20
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (C6A(-14+06))
<400> 20
uuugaccuac auguggaaag 20
<210> 21
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (C6A(-14+12))
<400> 21
uacauuuuug accuacaugu ggaaag 26
<210> 22
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (C6A(-13+09))
<400> 22
auuuuugacc uacaugggaa ag 22
<210> 23
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (CH6A(+69+91))
<400> 23
uacgaguuga uugucggacc cag 23
<210> 24
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (C6D(+12-13))
<400> 24
guggucuccu uaccuaugac ugugg 25
<210> 25
<211> 17
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (C6D(+06-11))
<400> 25
ggucuccuua ccuauga 17
<210> 26
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H6D(+04-21))
<400> 26
ugucucagua aucuucuuac cuau 24
<210> 27
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H6D(+18-04))
<400> 27
ucuuaccuau gacuauggau gaga 24
<210> 28
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H4A(+13+32))
<400> 28
gcaugaacuc uuguggaucc 20
<210> 29
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H4D(+04-16))
<400> 29
ccaggguacu acuuacauua 20
<210> 30
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H4D(-24-44))
<400> 30
aucguguguc acagcaucca g 21
<210> 31
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H4A(+11+40))
<400> 31
uguucagggc augaacucuu guggauccuu 30
<210> 32
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H3A(+30+60))
<400> 32
uaggaggcgc cucccauccu guaggucacu g 31
<210> 33
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H3A(+35+65))
<400> 33
aggucuagga ggcgccuccc auccuguagg u 31
<210> 34
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H3A(+30+54))
<400> 34
gcgccuccca uccuguaggu cacug 25
<210> 35
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H3D(+46-21))
<400> 35
cuucgaggag gucuaggagg cgccuc 26
<210> 36
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H3A(+30+50))
<400> 36
cucccauccu guaggucacu g 21
<210> 37
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H3D(+19-03))
<400> 37
uaccaguuuu ugcccuguca gg 22
<210> 38
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H3A(-06+20))
<400> 38
ucaauaugcu gcuucccaaa cugaaa 26
<210> 39
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H3A(+37+61))
<400> 39
cuaggaggcg ccucccaucc uguag 25
<210> 40
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H5A(+20+50))
<400> 40
uuaugauuuc caucuacgau gucaguacuu c 31
<210> 41
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H5D(+25-05))
<400> 41
cuuaccugcc aguggaggau uauauuccaa a 31
<210> 42
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H5D(+10-15))
<400> 42
caucaggauu cuuaccugcc agugg 25
<210> 43
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H5A(+10+34))
<400> 43
cgaugucagu acuuccaaua uucac 25
<210> 44
<211> 18
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H5D(-04-21))
<400> 44
accauucauc aggauucu 18
<210> 45
<211> 18
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H5D(+16-02))
<400> 45
accugccagu ggaggauu 18
<210> 46
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H5A(-07+20))
<400> 46
ccaauauuca cuaaaucaac cuguuaa 27
<210> 47
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H5D(+18-12))
<400> 47
caggauuguu accugccagu ggaggauuau 30
<210> 48
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H5A(+05+35))
<400> 48
acgaugucag uacuuccaau auucacuaaa u 31
<210> 49
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H5A(+15+45))
<400> 49
auuuccaucu acgaugucag uacuuccaau a 31
<210> 50
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H10A(-05+16))
<400> 50
caggagcuuc caaaugcugc a 21
<210> 51
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H10A(-05+24))
<400> 51
cuugucuuca ggagcuucca aaugcugca 29
<210> 52
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H10A(+98+119))
<400> 52
uccucagcag aaagaagcca cg 22
<210> 53
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H10A(+130+149))
<400> 53
uuagaaaucu cuccuugugc 20
<210> 54
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H10A(-33-14))
<400> 54
uaaauugggu guuacacaau 20
<210> 55
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H11D(+26+49))
<400> 55
cccugaggca uucccaucuu gaau 24
<210> 56
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H11D(+11-09))
<400> 56
aggacuuacu ugcuuuguuu 20
<210> 57
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H11A(+118+140))
<400> 57
cuugaauuua ggagauucau cug 23
<210> 58
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H11A(+75+97))
<400> 58
caucuucuga uaauuuuccu guu 23
<210> 59
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H12A(+52+75))
<400> 59
ucuucuguuu uuguuagcca guca 24
<210> 60
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H12A(-10+10))
<400> 60
ucuauguaaa cugaaaauuu 20
<210> 61
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H12A(+11+30))
<400> 61
uucuggagau ccauuaaaac 20
<210> 62
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H13A(+77+100))
<400> 62
cagcaguugc gugaucucca cuag 24
<210> 63
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H13A(+55+75))
<400> 63
uucaucaacu accaccacca u 21
<210> 64
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H13D(+06-19))
<400> 64
cuaagcaaaa uaaucugacc uuaag 25
<210> 65
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H14A(+37+64))
<400> 65
cuuguaaaag aacccagcgg ucuucugu 28
<210> 66
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H14A(+14+35))
<400> 66
caucuacaga uguuugccca uc 22
<210> 67
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H14A(+51+73))
<400> 67
gaaggauguc uuguaaaaga acc 23
<210> 68
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H14D(-02+18))
<400> 68
accuguucuu caguaagacg 20
<210> 69
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H14D(+14-10))
<400> 69
caugacacac cuguucuuca guaa 24
<210> 70
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H14A(+61+80))
<400> 70
cauuugagaa ggaugucuug 20
<210> 71
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H14A(-12+12))
<400> 71
aucucccaau accuggagaa gaga 24
<210> 72
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H15A(-12+19))
<400> 72
gccaugcacu aaaaaggcac ugcaagacau u 31
<210> 73
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H15A(+48+71))
<400> 73
ucuuuaaagc caguugugug aauc 24
<210> 74
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H15A(+08+28))
<400> 74
uuucugaaag ccaugcacua a 21
<210> 75
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H15D(+17-08))
<400> 75
guacauacgg ccaguuuuug aagac 25
<210> 76
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H16A(-12+19))
<400> 76
cuagauccgc uuuuaaaacc uguuaaaaca a 31
<210> 77
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H16A(-06+25))
<400> 77
ucuuuucuag auccgcuuuu aaaaccuguu a 31
<210> 78
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H16A(-06+19))
<400> 78
cuagauccgc uuuuaaaacc uguua 25
<210> 79
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H16A(+87+109))
<400> 79
ccgucuucug ggucacugac uua 23
<210> 80
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H16A(-07+19))
<400> 80
cuagauccgc uuuuaaaacc uguuaa 26
<210> 81
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H16A(-07+13))
<400> 81
ccgcuuuuaa aaccuguuaa 20
<210> 82
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H16A(+12+37))
<400> 82
uggauugcuu uuucuuuucu agaucc 26
<210> 83
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H16A(+92+116))
<400> 83
caugcuuccg ucuucugggu cacug 25
<210> 84
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H16A(+45+67))
<400> 84
gaucuuguuu gagugaauac agu 23
<210> 85
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H16A(+105+126))
<400> 85
guuauccagc caugcuuccg uc 22
<210> 86
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H16D(+05-20))
<400> 86
ugauaauugg uaucacuaac cugug 25
<210> 87
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H16D(+12-11))
<400> 87
guaucacuaa ccugugcugu ac 22
<210> 88
<211> 19
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H19A(+35+53))
<400> 88
cugcuggcau cuugcaguu 19
<210> 89
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H19A(+35+65))
<400> 89
gccugagcug aucugcuggc aucuugcagu u 31
<210> 90
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H20A(+44+71))
<400> 90
cuggcagaau ucgauccacc ggcuguuc 28
<210> 91
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H20A(+147+168))
<400> 91
cagcaguagu ugucaucugc uc 22
<210> 92
<211> 19
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H20A(+185+203))
<400> 92
ugauggggug guggguugg 19
<210> 93
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H20A(-08+17))
<400> 93
aucugcauua acacccucua gaaag 25
<210> 94
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H20A(+30+53))
<400> 94
ccggcuguuc aguuguucug aggc 24
<210> 95
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H20A(-11+17))
<400> 95
aucugcauua acacccucua gaaagaaa 28
<210> 96
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H20D(+08-20))
<400> 96
gaaggagaag agauucuuac cuuacaaa 28
<210> 97
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H20A(+44+63))
<400> 97
auucgaucca ccggcuguuc 20
<210> 98
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H20A(+149+168))
<400> 98
cagcaguagu ugucaucugc 20
<210> 99
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H21A(-06+16))
<400> 99
gccgguugac uucauccugu gc 22
<210> 100
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H21A(+85+106))
<400> 100
cugcauccag gaacaugggu cc 22
<210> 101
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H21A(+85+108))
<400> 101
gucugcaucc aggaacaugg guc 23
<210> 102
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H21A(+08+31))
<400> 102
guugaagauc ugauagccgg uuga 24
<210> 103
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H21D(+18-07))
<400> 103
uacuuacugu cuguagcucu uucu 24
<210> 104
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H22A(+22+45))
<400> 104
cacucauggu cuccugauag cgca 24
<210> 105
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H22A(+125+146))
<400> 105
cugcaauucc ccgagucucu gc 22
<210> 106
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H22A(+47+69))
<400> 106
acugcuggac ccauguccug aug 23
<210> 107
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H22A(+80+101))
<400> 107
cuaaguugag guauggagag u 21
<210> 108
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H22D(+13-11))
<400> 108
uauucacaga ccugcaauuc ccc 23
<210> 109
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H23A(+34+59))
<400> 109
acaguggugc ugagauagua uaggcc 26
<210> 110
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H23A(+18+39))
<400> 110
uaggccacuu uguugcucuu gc 22
<210> 111
<211> 19
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H23A(+72+90))
<400> 111
uucagagggc gcuuucuuc 19
<210> 112
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H24A(+48+70))
<400> 112
gggcaggcca uuccuccuuc aga 23
<210> 113
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H24A(-02+22))
<400> 113
ucuucagggu uuguauguga uucu 24
<210> 114
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H25A(+9+36))
<400> 114
cugggcugaa uugucugaau aucacug 27
<210> 115
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H25A(+131+156))
<400> 115
cuguuggcac augugauccc acugag 26
<210> 116
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H25D(+16-08))
<400> 116
gucuauaccu guuggcacau guga 24
<210> 117
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H26A(+132+156))
<400> 117
ugcuuucugu aauucaucug gaguu 25
<210> 118
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H26A(-07+19))
<400> 118
ccuccuuucu ggcauagacc uuccac 26
<210> 119
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H26A(+68+92))
<400> 119
ugugucaucc auucgugcau cucug 25
<210> 120
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H27A(+82+106))
<400> 120
uuaaggccuc uugugcuaca ggugg 25
<210> 121
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H27A(-4+19))
<400> 121
ggggcucuuc uuuagcucuc uga 23
<210> 122
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H27D(+19-03))
<400> 122
gacuuccaaa gucuugcauu uc 22
<210> 123
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H28A(-05+19))
<400> 123
gccaacaugc ccaaacuucc uaag 24
<210> 124
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H28A(+99+124))
<400> 124
cagagauuuc cucagcuccg ccagga 26
<210> 125
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H28D(+16-05))
<400> 125
cuuacaucua gcaccucaga g 21
<210> 126
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H29A(+57+81))
<400> 126
uccgccaucu guuagggucu gugcc 25
<210> 127
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H29A(+18+42))
<400> 127
auuuggguua uccucugaau gucgc 25
<210> 128
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H29D(+17-05))
<400> 128
cauaccucuu cauguaguuc cc 22
<210> 129
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H30A(+122+147))
<400> 129
cauuugagcu gcguccaccu ugucug 26
<210> 130
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H30A(+25+50))
<400> 130
uccugggcag acuggaugcu cuguuc 26
<210> 131
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H30D(+19-04))
<400> 131
uugccugggc uuccugaggc auu 23
<210> 132
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H31D(+06-18))
<400> 132
uucugaaaua acauauaccu gugc 24
<210> 133
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H31D(+03-22))
<400> 133
uaguuucuga aauaacauau accug 25
<210> 134
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H31A(+05+25))
<400> 134
gacuugucaa aucagauugg a 21
<210> 135
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H31D(+04-20))
<400> 135
guuucugaaa uaacauauac cugu 24
<210> 136
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H32D(+04-16))
<400> 136
caccagaaau acauaccaca 20
<210> 137
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H32A(+151+170))
<400> 137
caaugauuua gcugugacug 20
<210> 138
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H32A(+10+32))
<400> 138
cgaaacuuca uggagacauc uug 23
<210> 139
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H32A(+49+73))
<400> 139
cuuguagacg cugcucaaaa uuggc 25
<210> 140
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H33D(+09-11))
<400> 140
caugcacaca ccuuugcucc 20
<210> 141
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H33A(+53+76))
<400> 141
ucuguacaau cugacgucca gucu 24
<210> 142
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H33A(+30+56))
<400> 142
gucuuuauca ccauuuccac uucagac 27
<210> 143
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H33A(+64+88))
<400> 143
ccgucugcuu uuucuguaca aucug 25
<210> 144
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H34A(+83+104))
<400> 144
uccauaucug uagcugccag cc 22
<210> 145
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H34A(+143+165))
<400> 145
ccaggcaacu ucagaaucca aau 23
<210> 146
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H34A(-20+10))
<400> 146
uuucuguuac cugaaaagaa uuauaaugaa 30
<210> 147
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H34A(+46+70))
<400> 147
cauucauuuc cuuucgcauc uuacg 25
<210> 148
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H34A(+95+120))
<400> 148
ugaucucuuu gucaauucca uaucug 26
<210> 149
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H34D(+10-20))
<400> 149
uucagugaua uagguuuuac cuuuccccag 30
<210> 150
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H34A(+72+96))
<400> 150
cuguagcugc cagccauucu gucaag 26
<210> 151
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H35A(+141+161))
<400> 151
ucuucugcuc gggaggugac a 21
<210> 152
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H35A(+116+135))
<400> 152
ccaguuacua uucagaagac 20
<210> 153
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H35A(+24+43))
<400> 153
ucuucaggug caccuucugu 20
<210> 154
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H36A(+26+50))
<400> 154
ugugaugugg uccacauucu gguca 25
<210> 155
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H36A(-02+18))
<400> 155
ccauguguuu cugguauucc 20
<210> 156
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H37A(+26+50))
<400> 156
cguguagagu ccaccuuugg gcgua 25
<210> 157
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H37A(+82+105))
<400> 157
uacuaauuuc cugcaguggu cacc 24
<210> 158
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H37A(+134+157))
<400> 158
uucuguguga aauggcugca aauc 24
<210> 159
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H38A(-01+19))
<400> 159
ccuucaaagg aauggaggcc 20
<210> 160
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H38A(+59+83))
<400> 160
ugcugaauuu cagccuccag ugguu 25
<210> 161
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H38A(+88+112))
<400> 161
ugaagucuuc cucuuucaga uucac 25
<210> 162
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H39A(+62+85))
<400> 162
cuggcuuucu cucaucugug auuc 24
<210> 163
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H39A(+39+58))
<400> 163
guuguaaguu gucuccucuu 20
<210> 164
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H39A(+102+121))
<400> 164
uugucuguaa cagcugcugu 20
<210> 165
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H39D(+10-10))
<400> 165
gcucuaauac cuugagagca 20
<210> 166
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H40A(-05+17))
<400> 166
cuuugagacc ucaaauccug uu 22
<210> 167
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H40A(+129+153))
<400> 167
cuuuauuuuc cuuucaucuc ugggc 25
<210> 168
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H42A(-04+23))
<400> 168
aucguuucuu cacggacagu gugcugg 27
<210> 169
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H42A(+86+109))
<400> 169
gggcuuguga gacaugagug auuu 24
<210> 170
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H42D(+19-02))
<400> 170
accuucagag gacuccucuu gc 22
<210> 171
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H43D(+10-15))
<400> 171
uauguguuac cuacccuugu cgguc 25
<210> 172
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H43A(+101+120))
<400> 172
ggagagagcu uccuguagcu 20
<210> 173
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H43A(+78+100))
<400> 173
ucacccuuuc cacaggcguu gca 23
<210> 174
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H44A(+85+104))
<400> 174
uuugugucuu ucugagaaac 20
<210> 175
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H44D(+10-10))
<400> 175
aaagacuuac cuuaagauac 20
<210> 176
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H44A(-06+14))
<400> 176
aucugucaaa ucgccugcag 20
<210> 177
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H46D(+16-04))
<400> 177
uuaccuugac uugcucaagc 20
<210> 178
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H46A(+90+109))
<400> 178
uccagguuca agugggauac 20
<210> 179
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H47A(+76+100))
<400> 179
gcucuucugg gcuuauggga gcacu 25
<210> 180
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H47D(+25-02))
<400> 180
accuuuaucc acuggagauu ugucugc 27
<210> 181
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H47A(-9+12))
<400> 181
uuccaccagu aacugaaaca g 21
<210> 182
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H50A(+02+30))
<400> 182
ccacucagag cucagaucuu cuaacuucc 29
<210> 183
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H50A(+07+33))
<400> 183
cuuccacuca gagcucagau cuucuaa 27
<210> 184
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H51A(-01+25))
<400> 184
accagaguaa cagucugagu aggagc 26
<210> 185
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H51D(+16-07))
<400> 185
cucauaccuu cugcuugaug auc 23
<210> 186
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H51A(+111 +134))
<400> 186
uucuguccaa gcccgguuga aauc 24
<210> 187
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H51A(+66+95))
<400> 187
cuccaacauc aaggaagaug gcauuucuag 30
<210> 188
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H51D(+08-17))
<400> 188
aucauuuuuu cucauaccuu cugcu 25
<210> 189
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H51A/D(+08-17)& (-15+))
<400> 189
aucauuuuuu cucauaccuu cugcuaggag cuaaaa 36
<210> 190
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H51A(+175+195))
<400> 190
cacccaccau cacccucugu g 21
<210> 191
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H51A(+199+220))
<400> 191
aucaucucgu ugauauccuc aa 22
<210> 192
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H52A(-07+14))
<400> 192
uccugcauug uugccuguaa g 21
<210> 193
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H52A(+12+41))
<400> 193
uccaacuggg gacgccucug uuccaaaucc 30
<210> 194
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H52A(+17+37))
<400> 194
acuggggacg ccucuguucc a 21
<210> 195
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H52A(+93+112))
<400> 195
ccguaaugau uguucuagcc 20
<210> 196
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H52D(+05-15))
<400> 196
uguuaaaaaa cuuacuucga 20
<210> 197
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+45+69))
<400> 197
cauucaacug uugccuccgg uucug 25
<210> 198
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+39+62))
<400> 198
cuguugccuc cgguucugaa ggug 24
<210> 199
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+39+69))
<400> 199
cauucaacug uugccuccgg uucugaaggu g 31
<210> 200
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53D(+14-07))
<400> 200
uacuaaccuu gguuucugug a 21
<210> 201
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+23+47))
<400> 201
cugaaggugu ucuuguacuu caucc 25
<210> 202
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+150+176))
<400> 202
uguauaggga cccuccuucc augacuc 27
<210> 203
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53D(+20-05))
<400> 203
cuaaccuugg uuucugugau uuucu 25
<210> 204
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53D(+09-18))
<400> 204
gguaucuuug auacuaaccu ugguuuc 27
<210> 205
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(-12+10))
<400> 205
auucuuucaa cuagaauaaa ag 22
<210> 206
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(-07+18))
<400> 206
gauucugaau ucuuucaacu agaau 25
<210> 207
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+07+26))
<400> 207
aucccacuga uucugaauuc 20
<210> 208
<211> 22
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+124+145))
<400> 208
uuggcucugg ccuguccuaa ga 22
<210> 209
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H46A(+86+115))
<400> 209
cucuuuucca gguucaagug ggauacuagc 30
<210> 210
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H46A(+107+137))
<400> 210
caagcuuuuc uuuuaguugc ugcucuuuuc c 31
<210> 211
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H46A(-10+20))
<400> 211
uauucuuuug uucuucuagc cuggagaaag 30
<210> 212
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H46A(+50+77))
<400> 212
cugcuuccuc caaccauaaa acaaauuc 28
<210> 213
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H45A(-06+20))
<400> 213
ccaaugccau ccuggaguuc cuguaa 26
<210> 214
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H45A(+91 +110))
<400> 214
uccuguagaa uacuggcauc 20
<210> 215
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H45A(+125+151))
<400> 215
ugcagaccuc cugccaccgc agauuca 27
<210> 216
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H45D(+16 -04))
<400> 216
cuaccucuuu uuucugucug 20
<210> 217
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic (H45A(+71+90))
<400> 217
uguuuuugag gauugcugaa 20
<210> 218
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+33+60))
<400> 218
gttgcctccg gttctgaagg tgttcttg 28
<210> 219
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+23+47))
<400> 219
ctgaaggtgt tcttgtactt catcc 25
<210> 220
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+33+62))
<400> 220
ctgttgcctc cggttctgaa ggtgttcttg 30
<210> 221
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+33+65))
<400> 221
caactgttgc ctccggttct gaaggtgttc ttg 33
<210> 222
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+31+55))
<400> 222
ctccggttct gaaggtgttc ttgta 25
<210> 223
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+46+73))
<400> 223
atttcattca actgttgcct ccggttct 28
<210> 224
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+22+46))
<400> 224
tgaaggtgtt cttgtacttc atccc 25
<210> 225
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+46+69))
<400> 225
cattcaactg ttgcctccgg ttct 24
<210> 226
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+40+61))
<400> 226
tgttgcctcc ggttctgaag gt 22
<210> 227
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+30+60))
<400> 227
gttgcctccg gttctgaagg tgttc 25
<210> 228
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+30+57))
<400> 228
gcctccggtt ctgaaggtgt tcttgtac 28
<210> 229
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+30+56))
<400> 229
cctccggttc tgaaggtgtt cttgtac 27
<210> 230
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+30+55))
<400> 230
ctccggttct gaaggtgttc ttgtac 26
<210> 231
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+33+57))
<400> 231
gcctccggtt ctgaaggtgt tcttg 25
<210> 232
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H44A(-07+17))
<400> 232
cagatctgtc aaatcgcctg cagg 24
<210> 233
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H44A(-07+20))
<400> 233
caacagatct gtcaaatcgc ctgcagg 27
<210> 234
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H44A(-07+22))
<400> 234
ctcaacagat ctgtcaaatc gcctgcagg 29
<210> 235
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H44A(+77+101))
<400> 235
gtgtctttct gagaaactgt tcagc 25
<210> 236
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H44A(+64+91))
<400> 236
gagaaactgt tcagcttctg ttagccac 28
<210> 237
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H44A(+62+89))
<400> 237
gaaactgttc agcttctgtt agccactg 28
<210> 238
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H44A(+62+85))
<400> 238
ctgttcagct tctgttagcc actg 24
<210> 239
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H44A(-06+14))
<400> 239
atctgtcaaa tcgcctgcag 20
<210> 240
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H44A(+85+104))
<400> 240
tttgtgtctt tctgagaaac 20
<210> 241
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H44A(+61+84))
<400> 241
tgttcagctt ctgttagcca ctga 24
<210> 242
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H44A(-10+15))
<400> 242
gatctgtcaa atcgcctgca ggtaa 25
<210> 243
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H44A(+64+88))
<400> 243
aaactgttca gcttctgtta gccac 25
<210> 244
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H44A(+79+103))
<400> 244
ttgtgtcttt ctgagaaact gttca 25
<210> 245
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H44A(-06+20))
<400> 245
caacagatct gtcaaatcgc ctgcag 26
<210> 246
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H44A(-09+17))
<400> 246
cagatctgtc aaatcgcctg caggta 26
<210> 247
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H44A(+59+85))
<400> 247
ctgttcagct tctgttagcc actgatt 27
<210> 248
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H44A(+59+89))
<400> 248
gaaactgttc agcttctgtt agccactgat t 31
<210> 249
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H44A(+65+90))
<400> 249
agaaactgtt cagcttctgt tagcca 26
<210> 250
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon44.25.001)
<400> 250
ctgcaggtaa aagcatatgg atcaa 25
<210> 251
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon44.25.002)
<400> 251
atcgcctgca ggtaaaagca tatgg 25
<210> 252
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon44.25.003)
<400> 252
gtcaaatcgc ctgcaggtaa aagca 25
<210> 253
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon44.25.005)
<400> 253
caacagatct gtcaaatcgc ctgca 25
<210> 254
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon44.25.006)
<400> 254
tttctcaaca gatctgtcaa atcgc 25
<210> 255
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon44.25.007)
<400> 255
ccatttctca acagatctgt caaat 25
<210> 256
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon44.25.008)
<400> 256
ataatgaaaa cgccgccatt tctca 25
<210> 257
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon44.25.009)
<400> 257
aaatatcttt atatcataat gaaaa 25
<210> 258
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon44.25.010)
<400> 258
tgttagccac tgattaaata tcttt 25
<210> 259
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon44.25.013)
<400> 259
ccaattctca ggaatttgtg tcttt 25
<210> 260
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon44.25.014)
<400> 260
gtatttagca tgttcccaat tctca 25
<210> 261
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon44.25.015)
<400> 261
cttaagatac catttgtatt tagca 25
<210> 262
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon44.25.016)
<400> 262
cttaccttaa gataccattt gtatt 25
<210> 263
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon44.25.017)
<400> 263
aaagacttac cttaagatac cattt 25
<210> 264
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon44.25.018)
<400> 264
aaatcaaaga cttaccttaa gatac 25
<210> 265
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon44.25.019)
<400> 265
aaaacaaatc aaagacttac cttaa 25
<210> 266
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon44.25.020)
<400> 266
tcgaaaaaac aaatcaaaga cttac 25
<210> 267
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.001)
<400> 267
ctgtaagata ccaaaaaggc aaaac 25
<210> 268
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.002)
<400> 268
cctgtaagat accaaaaagg caaaa 25
<210> 269
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.002.2)
<400> 269
agttcctgta agataccaaa aaggc 25
<210> 270
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.003)
<400> 270
gagttcctgt aagataccaa aaagg 25
<210> 271
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.003.2)
<400> 271
cctggagttc ctgtaagata ccaaa 25
<210> 272
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.004)
<400> 272
tcctggagtt cctgtaagat accaa 25
<210> 273
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.004.2)
<400> 273
gccatcctgg agttcctgta agata 25
<210> 274
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.005)
<400> 274
tgccatcctg gagttcctgt aagat 25
<210> 275
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.005.2)
<400> 275
ccaatgccat cctggagttc ctgta 25
<210> 276
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.006)
<400> 276
cccaatgcca tcctggagtt cctgt 25
<210> 277
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.006.2)
<400> 277
gctgcccaat gccatcctgg agttc 25
<210> 278
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.007)
<400> 278
cgctgcccaa tgccatcctg gagtt 25
<210> 279
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.008)
<400> 279
aacagtttgc cgctgcccaa tgcca 25
<210> 280
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.008.2)
<400> 280
ctgacaacag tttgccgctg cccaa 25
<210> 281
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.009)
<400> 281
gttgcattca atgttctgac aacag 25
<210> 282
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.010)
<400> 282
gctgaattat ttcttcccca gttgc 25
<210> 283
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.010.2)
<400> 283
attatttctt ccccagttgc attca 25
<210> 284
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.011)
<400> 284
ggcatctgtt tttgaggatt gctga 25
<210> 285
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.011.2)
<400> 285
tttgaggatt gctgaattat ttctt 25
<210> 286
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.012)
<400> 286
aatttttcct gtagaatact ggcat 25
<210> 287
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.012.2)
<400> 287
atactggcat ctgtttttga ggatt 25
<210> 288
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.013)
<400> 288
accgcagatt caggcttccc aattt 25
<210> 289
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.014)
<400> 289
ctgtttgcag acctcctgcc accgc 25
<210> 290
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.014.2)
<400> 290
agattcaggc ttcccaattt ttcct 25
<210> 291
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.015)
<400> 291
ctcttttttc tgtctgacag ctgtt 25
<210> 292
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.015.2)
<400> 292
acctcctgcc accgcagatt caggc 25
<210> 293
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.016)
<400> 293
cctacctctt ttttctgtct gacag 25
<210> 294
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.016.2)
<400> 294
gacagctgtt tgcagacctc ctgcc 25
<210> 295
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.017)
<400> 295
gtcgccctac ctcttttttc tgtct 25
<210> 296
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.018)
<400> 296
gatctgtcgc cctacctctt ttttc 25
<210> 297
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.019)
<400> 297
tattagatct gtcgccctac ctctt 25
<210> 298
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.25.020)
<400> 298
attcctatta gatctgtcgc cctac 25
<210> 299
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.001)
<400> 299
agataccaaa aaggcaaaac 20
<210> 300
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.002)
<400> 300
aagataccaa aaaggcaaaa 20
<210> 301
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.003)
<400> 301
cctgtaagat accaaaaagg 20
<210> 302
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.004)
<400> 302
gagttcctgt aagataccaa 20
<210> 303
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.005)
<400> 303
tcctggagtt cctgtaagat 20
<210> 304
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.006)
<400> 304
tgccatcctg gagttcctgt 20
<210> 305
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.007)
<400> 305
cccaatgcca tcctggagtt 20
<210> 306
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.008)
<400> 306
cgctgcccaa tgccatcctg 20
<210> 307
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.009)
<400> 307
ctgacaacag tttgccgctg 20
<210> 308
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.010)
<400> 308
gttgcattca atgttctgac 20
<210> 309
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.011)
<400> 309
attatttctt ccccagttgc 20
<210> 310
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.012)
<400> 310
tttgaggatt gctgaattat 20
<210> 311
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.013)
<400> 311
atactggcat ctgtttttga 20
<210> 312
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.014)
<400> 312
aatttttcct gtagaatact 20
<210> 313
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.015)
<400> 313
agattcaggc ttcccaattt 20
<210> 314
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.016)
<400> 314
acctcctgcc accgcagatt 20
<210> 315
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.017)
<400> 315
gacagctgtt tgcagacctc 20
<210> 316
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.018)
<400> 316
ctcttttttc tgtctgacag 20
<210> 317
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.019)
<400> 317
cctacctctt ttttctgtct 20
<210> 318
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.020)
<400> 318
gtcgccctac ctcttttttc 20
<210> 319
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.021)
<400> 319
gatctgtcgc cctacctctt 20
<210> 320
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.022)
<400> 320
tattagatct gtcgccctac 20
<210> 321
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon45.20.023)
<400> 321
attcctatta gatctgtcgc 20
<210> 322
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.001)
<400> 322
gggggatttg agaaaataaa attac 25
<210> 323
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.002)
<400> 323
atttgagaaa ataaaattac cttga 25
<210> 324
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.002.2)
<400> 324
ctagcctgga gaaagaagaa taaaa 25
<210> 325
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.003)
<400> 325
agaaaataaa attaccttga cttgc 25
<210> 326
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.003.2)
<400> 326
ttcttctagc ctggagaaag aagaa 25
<210> 327
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.004)
<400> 327
ataaaattac cttgacttgc tcaag 25
<210> 328
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.004.2)
<400> 328
ttttgttctt ctagcctgga gaaag 25
<210> 329
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.005)
<400> 329
attaccttga cttgctcaag ctttt 25
<210> 330
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.005.2)
<400> 330
tattcttttg ttcttctagc ctgga 25
<210> 331
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.006)
<400> 331
cttgacttgc tcaagctttt ctttt 25
<210> 332
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.006.2)
<400> 332
caagatattc ttttgttctt ctagc 25
<210> 333
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.007)
<400> 333
cttttagttg ctgctctttt ccagg 25
<210> 334
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.008)
<400> 334
ccaggttcaa gtgggatact agcaa 25
<210> 335
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.008.2)
<400> 335
atctctttga aattctgaca agata 25
<210> 336
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.009)
<400> 336
agcaatgtta tctgcttcct ccaac 25
<210> 337
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.009.2)
<400> 337
aacaaattca tttaaatctc tttga 25
<210> 338
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.010)
<400> 338
ccaaccataa aacaaattca tttaa 25
<210> 339
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.010.2)
<400> 339
ttcctccaac cataaaacaa attca 25
<210> 340
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.011)
<400> 340
tttaaatctc tttgaaattc tgaca 25
<210> 341
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.012)
<400> 341
tgacaagata ttcttttgtt cttct 25
<210> 342
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.012.2)
<400> 342
ttcaagtggg atactagcaa tgtta 25
<210> 343
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.013)
<400> 343
agatattctt ttgttcttct agcct 25
<210> 344
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.013.2)
<400> 344
ctgctctttt ccaggttcaa gtggg 25
<210> 345
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.014)
<400> 345
ttcttttgtt cttctagcct ggaga 25
<210> 346
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.014.2)
<400> 346
cttttctttt agttgctgct ctttt 25
<210> 347
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.015)
<400> 347
ttgttcttct agcctggaga aagaa 25
<210> 348
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.016)
<400> 348
cttctagcct ggagaaagaa gaata 25
<210> 349
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.017)
<400> 349
agcctggaga aagaagaata aaatt 25
<210> 350
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.25.018)
<400> 350
ctggagaaag aagaataaaa ttgtt 25
<210> 351
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.20.001)
<400> 351
gaaagaagaa taaaattgtt 20
<210> 352
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.20.002)
<400> 352
ggagaaagaa gaataaaatt 20
<210> 353
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.20.003)
<400> 353
agcctggaga aagaagaata 20
<210> 354
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.20.004)
<400> 354
cttctagcct ggagaaagaa 20
<210> 355
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.20.005)
<400> 355
ttgttcttct agcctggaga 20
<210> 356
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.20.006)
<400> 356
ttcttttgtt cttctagcct 20
<210> 357
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.20.007)
<400> 357
tgacaagata ttcttttgtt 20
<210> 358
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.20.008)
<400> 358
atctctttga aattctgaca 20
<210> 359
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.20.009)
<400> 359
aacaaattca tttaaatctc 20
<210> 360
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.20.010)
<400> 360
ttcctccaac cataaaacaa 20
<210> 361
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.20.011)
<400> 361
agcaatgtta tctgcttcct 20
<210> 362
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.20.012)
<400> 362
ttcaagtggg atactagcaa 20
<210> 363
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.20.013)
<400> 363
ctgctctttt ccaggttcaa 20
<210> 364
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.20.014)
<400> 364
cttttctttt agttgctgct 20
<210> 365
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.20.015)
<400> 365
cttgacttgc tcaagctttt 20
<210> 366
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.20.016)
<400> 366
attaccttga cttgctcaag 20
<210> 367
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.20.017)
<400> 367
ataaaattac cttgacttgc 20
<210> 368
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.20.018)
<400> 368
agaaaataaa attaccttga 20
<210> 369
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.20.019)
<400> 369
atttgagaaa ataaaattac 20
<210> 370
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon46.20.020)
<400> 370
gggggatttg agaaaataaa 20
<210> 371
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.001)
<400> 371
ctgaaacaga caaatgcaac aacgt 25
<210> 372
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.002)
<400> 372
agtaactgaa acagacaaat gcaac 25
<210> 373
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.003)
<400> 373
ccaccagtaa ctgaaacaga caaat 25
<210> 374
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.004)
<400> 374
ctcttccacc agtaactgaa acaga 25
<210> 375
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.005)
<400> 375
ggcaactctt ccaccagtaa ctgaa 25
<210> 376
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.006)
<400> 376
gcaggggcaa ctcttccacc agtaa 25
<210> 377
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.007)
<400> 377
ctggcgcagg ggcaactctt ccacc 25
<210> 378
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.008)
<400> 378
tttaattgtt tgagaattcc ctggc 25
<210> 379
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.008.2)
<400> 379
ttgtttgaga attccctggc gcagg 25
<210> 380
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.009)
<400> 380
gcacgggtcc tccagtttca tttaa 25
<210> 381
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.009.2)
<400> 381
tccagtttca tttaattgtt tgaga 25
<210> 382
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.010)
<400> 382
gcttatggga gcacttacaa gcacg 25
<210> 383
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.010.2)
<400> 383
tacaagcacg ggtcctccag tttca 25
<210> 384
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.011)
<400> 384
agtttatctt gctcttctgg gctta 25
<210> 385
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.012)
<400> 385
tctgcttgag cttattttca agttt 25
<210> 386
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.012.2)
<400> 386
atcttgctct tctgggctta tggga 25
<210> 387
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.013)
<400> 387
ctttatccac tggagatttg tctgc 25
<210> 388
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.013.2)
<400> 388
cttattttca agtttatctt gctct 25
<210> 389
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.014)
<400> 389
ctaaccttta tccactggag atttg 25
<210> 390
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.014.2)
<400> 390
atttgtctgc ttgagcttat tttca 25
<210> 391
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.015)
<400> 391
aatgtctaac ctttatccac tggag 25
<210> 392
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.016)
<400> 392
tggttaatgt ctaaccttta tccac 25
<210> 393
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.017)
<400> 393
agagatggtt aatgtctaac cttta 25
<210> 394
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.25.018)
<400> 394
acggaagaga tggttaatgt ctaac 25
<210> 395
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.20.001)
<400> 395
acagacaaat gcaacaacgt 20
<210> 396
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.20.002)
<400> 396
ctgaaacaga caaatgcaac 20
<210> 397
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.20.003)
<400> 397
agtaactgaa acagacaaat 20
<210> 398
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.20.004)
<400> 398
ccaccagtaa ctgaaacaga 20
<210> 399
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.20.005)
<400> 399
ctcttccacc agtaactgaa 20
<210> 400
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.20.006)
<400> 400
ggcaactctt ccaccagtaa 20
<210> 401
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.20.007)
<400> 401
ctggcgcagg ggcaactctt 20
<210> 402
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.20.008)
<400> 402
ttgtttgaga attccctggc 20
<210> 403
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.20.009)
<400> 403
tccagtttca tttaattgtt 20
<210> 404
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.20.010)
<400> 404
tacaagcacg ggtcctccag 20
<210> 405
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.20.011)
<400> 405
gcttatggga gcacttacaa 20
<210> 406
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.20.012)
<400> 406
atcttgctct tctgggctta 20
<210> 407
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.20.013)
<400> 407
cttattttca agtttatctt 20
<210> 408
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.20.014)
<400> 408
atttgtctgc ttgagcttat 20
<210> 409
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.20.015)
<400> 409
ctttatccac tggagatttg 20
<210> 410
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.20.016)
<400> 410
ctaaccttta tccactggag 20
<210> 411
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.20.017)
<400> 411
aatgtctaac ctttatccac 20
<210> 412
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.20.018)
<400> 412
tggttaatgt ctaaccttta 20
<210> 413
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.20.019)
<400> 413
agagatggtt aatgtctaac 20
<210> 414
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon47.20.020)
<400> 414
acggaagaga tggttaatgt 20
<210> 415
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.001)
<400> 415
ctgaaaggaa aatacatttt aaaaa 25
<210> 416
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.002)
<400> 416
cctgaaagga aaatacattt taaaa 25
<210> 417
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.002.2)
<400> 417
gaaacctgaa aggaaaatac atttt 25
<210> 418
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.003)
<400> 418
ggaaacctga aaggaaaata cattt 25
<210> 419
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.003.2)
<400> 419
ctctggaaac ctgaaaggaa aatac 25
<210> 420
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.004)
<400> 420
gctctggaaa cctgaaagga aaata 25
<210> 421
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.004.2)
<400> 421
taaagctctg gaaacctgaa aggaa 25
<210> 422
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.005)
<400> 422
gtaaagctct ggaaacctga aagga 25
<210> 423
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.005.2)
<400> 423
tcaggtaaag ctctggaaac ctgaa 25
<210> 424
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.006)
<400> 424
ctcaggtaaa gctctggaaa cctga 25
<210> 425
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.006.2)
<400> 425
gtttctcagg taaagctctg gaaac 25
<210> 426
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.007)
<400> 426
tgtttctcag gtaaagctct ggaaa 25
<210> 427
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.007.2)
<400> 427
aatttctcct tgtttctcag gtaaa 25
<210> 428
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.008)
<400> 428
tttgagcttc aatttctcct tgttt 25
<210> 429
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.008)
<400> 429
ttttatttga gcttcaattt ctcct 25
<210> 430
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.009)
<400> 430
aagctgccca aggtctttta tttga 25
<210> 431
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.010)
<400> 431
aggtcttcaa gctttttttc aagct 25
<210> 432
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.010.2)
<400> 432
ttcaagcttt ttttcaagct gccca 25
<210> 433
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.011)
<400> 433
gatgatttaa ctgctcttca aggtc 25
<210> 434
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.011.2)
<400> 434
ctgctcttca aggtcttcaa gcttt 25
<210> 435
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.012)
<400> 435
aggagataac cacagcagca gatga 25
<210> 436
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.012.2)
<400> 436
cagcagatga tttaactgct cttca 25
<210> 437
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.013)
<400> 437
atttccaact gattcctaat aggag 25
<210> 438
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.014)
<400> 438
cttggtttgg ttggttataa atttc 25
<210> 439
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.014.2)
<400> 439
caactgattc ctaataggag ataac 25
<210> 440
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.015)
<400> 440
cttaacgtca aatggtcctt cttgg 25
<210> 441
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.015.2)
<400> 441
ttggttataa atttccaact gattc 25
<210> 442
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.016)
<400> 442
cctaccttaa cgtcaaatgg tcctt 25
<210> 443
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.016.2)
<400> 443
tccttcttgg tttggttggt tataa 25
<210> 444
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.017)
<400> 444
agttccctac cttaacgtca aatgg 25
<210> 445
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.018)
<400> 445
caaaaagttc cctaccttaa cgtca 25
<210> 446
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.019)
<400> 446
taaagcaaaa agttccctac cttaa 25
<210> 447
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.25.020)
<400> 447
atatttaaag caaaaagttc cctac 25
<210> 448
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.001)
<400> 448
aggaaaatac attttaaaaa 20
<210> 449
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.002)
<400> 449
aaggaaaata cattttaaaa 20
<210> 450
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.003)
<400> 450
cctgaaagga aaatacattt 20
<210> 451
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.004)
<400> 451
ggaaacctga aaggaaaata 20
<210> 452
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.005)
<400> 452
gctctggaaa cctgaaagga 20
<210> 453
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.006)
<400> 453
gtaaagctct ggaaacctga 20
<210> 454
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.007)
<400> 454
ctcaggtaaa gctctggaaa 20
<210> 455
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.008)
<400> 455
aatttctcct tgtttctcag 20
<210> 456
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.009)
<400> 456
ttttatttga gcttcaattt 20
<210> 457
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.010)
<400> 457
aagctgccca aggtctttta 20
<210> 458
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.011)
<400> 458
ttcaagcttt ttttcaagct 20
<210> 459
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.012)
<400> 459
ctgctcttca aggtcttcaa 20
<210> 460
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.013)
<400> 460
cagcagatga tttaactgct 20
<210> 461
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.014)
<400> 461
aggagataac cacagcagca 20
<210> 462
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.015)
<400> 462
caactgattc ctaataggag 20
<210> 463
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.016)
<400> 463
ttggttataa atttccaact 20
<210> 464
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.017)
<400> 464
tccttcttgg tttggttggt 20
<210> 465
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.018)
<400> 465
cttaacgtca aatggtcctt 20
<210> 466
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.019)
<400> 466
cctaccttaa cgtcaaatgg 20
<210> 467
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.020)
<400> 467
agttccctac cttaacgtca 20
<210> 468
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.021)
<400> 468
caaaaagttc cctaccttaa 20
<210> 469
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.022)
<400> 469
taaagcaaaa agttccctac 20
<210> 470
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon48.20.023)
<400> 470
atatttaaag caaaaagttc 20
<210> 471
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.001)
<400> 471
ctggggaaaa gaacccatat agtgc 25
<210> 472
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.002)
<400> 472
tcctggggaa aagaacccat atagt 25
<210> 473
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.002.2)
<400> 473
gtttcctggg gaaaagaacc catat 25
<210> 474
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.003)
<400> 474
cagtttcctg gggaaaagaa cccat 25
<210> 475
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.003.2)
<400> 475
tttcagtttc ctggggaaaa gaacc 25
<210> 476
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.004)
<400> 476
tatttcagtt tcctggggaa aagaa 25
<210> 477
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.004.2)
<400> 477
tgctatttca gtttcctggg gaaaa 25
<210> 478
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.005)
<400> 478
actgctattt cagtttcctg gggaa 25
<210> 479
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.005.2)
<400> 479
tgaactgcta tttcagtttc ctggg 25
<210> 480
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.006)
<400> 480
cttgaactgc tatttcagtt tcctg 25
<210> 481
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.006.2)
<400> 481
tagcttgaac tgctatttca gtttc 25
<210> 482
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.007)
<400> 482
tttagcttga actgctattt cagtt 25
<210> 483
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.008)
<400> 483
ttccacatcc ggttgtttag cttga 25
<210> 484
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.009)
<400> 484
tgccctttag acaaaatctc ttcca 25
<210> 485
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.009.2)
<400> 485
tttagacaaa atctcttcca catcc 25
<210> 486
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.010)
<400> 486
gtttttcctt gtacaaatgc tgccc 25
<210> 487
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.010.2)
<400> 487
gtacaaatgc tgccctttag acaaa 25
<210> 488
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.011)
<400> 488
cttcactggc tgagtggctg gtttt 25
<210> 489
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.011.2)
<400> 489
ggctggtttt tccttgtaca aatgc 25
<210> 490
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.012)
<400> 490
attaccttca ctggctgagt ggctg 25
<210> 491
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.013)
<400> 491
gcttcattac cttcactggc tgagt 25
<210> 492
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.014)
<400> 492
aggttgcttc attaccttca ctggc 25
<210> 493
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.015)
<400> 493
gctagaggtt gcttcattac cttca 25
<210> 494
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.25.016)
<400> 494
atattgctag aggttgcttc attac 25
<210> 495
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.20.001)
<400> 495
gaaaagaacc catatagtgc 20
<210> 496
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.20.002)
<400> 496
gggaaaagaa cccatatagt 20
<210> 497
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.20.003)
<400> 497
tcctggggaa aagaacccat 20
<210> 498
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.20.004)
<400> 498
cagtttcctg gggaaaagaa 20
<210> 499
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.20.005)
<400> 499
tatttcagtt tcctggggaa 20
<210> 500
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.20.006)
<400> 500
actgctattt cagtttcctg 20
<210> 501
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.20.007)
<400> 501
cttgaactgc tatttcagtt 20
<210> 502
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.20.008)
<400> 502
tttagcttga actgctattt 20
<210> 503
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.20.009)
<400> 503
ttccacatcc ggttgtttag 20
<210> 504
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.20.010)
<400> 504
tttagacaaa atctcttcca 20
<210> 505
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.20.011)
<400> 505
gtacaaatgc tgccctttag 20
<210> 506
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.20.012)
<400> 506
ggctggtttt tccttgtaca 20
<210> 507
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.20.013)
<400> 507
cttcactggc tgagtggctg 20
<210> 508
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.20.014)
<400> 508
attaccttca ctggctgagt 20
<210> 509
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.20.015)
<400> 509
gcttcattac cttcactggc 20
<210> 510
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.20.016)
<400> 510
aggttgcttc attaccttca 20
<210> 511
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.20.017)
<400> 511
gctagaggtt gcttcattac 20
<210> 512
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon49.20.018)
<400> 512
atattgctag aggttgcttc 20
<210> 513
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.001)
<400> 513
ctttaacaga aaagcataca catta 25
<210> 514
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.002)
<400> 514
tcctctttaa cagaaaagca tacac 25
<210> 515
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.002.2)
<400> 515
ttcctcttta acagaaaagc ataca 25
<210> 516
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.003)
<400> 516
taacttcctc tttaacagaa aagca 25
<210> 517
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.003.2)
<400> 517
ctaacttcct ctttaacaga aaagc 25
<210> 518
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.004)
<400> 518
tcttctaact tcctctttaa cagaa 25
<210> 519
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.004.2)
<400> 519
atcttctaac ttcctcttta acaga 25
<210> 520
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.005)
<400> 520
tcagatcttc taacttcctc tttaa 25
<210> 521
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.005.2)
<400> 521
ctcagatctt ctaacttcct cttta 25
<210> 522
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.006)
<400> 522
agagctcaga tcttctaact tcctc 25
<210> 523
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.006.2 NG-08-0731)
<400> 523
cagagctcag atcttctaac ttcct 25
<210> 524
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.007)
<400> 524
cactcagagc tcagatcttc tact 24
<210> 525
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.007.2)
<400> 525
ccttccactc agagctcaga tcttc 25
<210> 526
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.008)
<400> 526
gtaaacggtt taccgccttc cactc 25
<210> 527
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.009)
<400> 527
ctttgccctc agctcttgaa gtaaa 25
<210> 528
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.009.2)
<400> 528
ccctcagctc ttgaagtaaa cggtt 25
<210> 529
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.010)
<400> 529
ccaggagcta ggtcaggctg ctttg 25
<210> 530
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.010.2)
<400> 530
ggtcaggctg ctttgccctc agctc 25
<210> 531
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.011)
<400> 531
aggctccaat agtggtcagt ccagg 25
<210> 532
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.011.2)
<400> 532
tcagtccagg agctaggtca ggctg 25
<210> 533
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.012 AVI-5038)
<400> 533
cttacaggct ccaatagtgg tcagt 25
<210> 534
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.013)
<400> 534
gtatacttac aggctccaat agtgg 25
<210> 535
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.014)
<400> 535
atccagtata cttacaggct ccaat 25
<210> 536
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.015 NG-08-0741)
<400> 536
atgggatcca gtatacttac aggct 25
<210> 537
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.25.016 NG-08-0742)
<400> 537
agagaatggg atccagtata cttac 25
<210> 538
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.20.001)
<400> 538
acagaaaagc atacacatta 20
<210> 539
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.20.002)
<400> 539
tttaacagaa aagcatacac 20
<210> 540
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.20.003)
<400> 540
tcctctttaa cagaaaagca 20
<210> 541
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.20.004)
<400> 541
taacttcctc tttaacagaa 20
<210> 542
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.20.005)
<400> 542
tcttctaact tcctctttaa 20
<210> 543
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.20.006)
<400> 543
tcagatcttc taacttcctc 20
<210> 544
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.20.007)
<400> 544
ccttccactc agagctcaga 20
<210> 545
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.20.008)
<400> 545
gtaaacggtt taccgccttc 20
<210> 546
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.20.009)
<400> 546
ccctcagctc ttgaagtaaa 20
<210> 547
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.20.010)
<400> 547
ggtcaggctg ctttgccctc 20
<210> 548
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.20.011)
<400> 548
tcagtccagg agctaggtca 20
<210> 549
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.20.012)
<400> 549
aggctccaat agtggtcagt 20
<210> 550
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.20.013)
<400> 550
cttacaggct ccaatagtgg 20
<210> 551
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.20.014)
<400> 551
gtatacttac aggctccaat 20
<210> 552
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.20.015)
<400> 552
atccagtata cttacaggct 20
<210> 553
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.20.016)
<400> 553
atgggatcca gtatacttac 20
<210> 554
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon50.20.017)
<400> 554
agagaatggg atccagtata 20
<210> 555
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.001-44)
<400> 555
ctaaaatatt ttgggttttt gcaaaa 26
<210> 556
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.002-45)
<400> 556
gctaaaatat tttgggtttt tgcaaa 26
<210> 557
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.002.2-46)
<400> 557
taggagctaa aatattttgg gttttt 26
<210> 558
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.003)
<400> 558
agtaggagct aaaatatttt gggtt 25
<210> 559
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.003.2)
<400> 559
tgagtaggag ctaaaatatt ttggg 25
<210> 560
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.004)
<400> 560
ctgagtagga gctaaaatat tttggg 26
<210> 561
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.004.2)
<400> 561
cagtctgagt aggagctaaa atatt 25
<210> 562
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.005)
<400> 562
acagtctgag taggagctaa aatatt 26
<210> 563
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.005.2)
<400> 563
gagtaacagt ctgagtagga gctaaa 26
<210> 564
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.006)
<400> 564
cagagtaaca gtctgagtag gagct 25
<210> 565
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.006.2)
<400> 565
caccagagta acagtctgag taggag 26
<210> 566
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.007)
<400> 566
gtcaccagag taacagtctg agtag 25
<210> 567
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.007.2)
<400> 567
aaccacaggt tgtgtcacca gagtaa 26
<210> 568
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.008)
<400> 568
gttgtgtcac cagagtaaca gtctg 25
<210> 569
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.009)
<400> 569
tggcagtttc cttagtaacc acaggt 26
<210> 570
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.010)
<400> 570
atttctagtt tggagatggc agtttc 26
<210> 571
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.010.2)
<400> 571
ggaagatggc atttctagtt tggag 25
<210> 572
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.011)
<400> 572
catcaaggaa gatggcattt ctagtt 26
<210> 573
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.011.2)
<400> 573
gagcaggtac ctccaacatc aaggaa 26
<210> 574
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.012)
<400> 574
atctgccaga gcaggtacct ccaac 25
<210> 575
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.013)
<400> 575
aagttctgtc caagcccggt tgaaat 26
<210> 576
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.013.2)
<400> 576
cggttgaaat ctgccagagc aggtac 26
<210> 577
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.014)
<400> 577
gagaaagcca gtcggtaagt tctgtc 26
<210> 578
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.014.2)
<400> 578
gtcggtaagt tctgtccaag cccgg 25
<210> 579
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.015)
<400> 579
ataacttgat caagcagaga aagcca 26
<210> 580
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.015.2)
<400> 580
aagcagagaa agccagtcgg taagt 25
<210> 581
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.016)
<400> 581
caccctctgt gattttataa cttgat 26
<210> 582
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.017)
<400> 582
caaggtcacc caccatcacc ctctgt 26
<210> 583
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.017.2)
<400> 583
catcaccctc tgtgatttta taact 25
<210> 584
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.018)
<400> 584
cttctgcttg atgatcatct cgttga 26
<210> 585
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.019)
<400> 585
ccttctgctt gatgatcatc tcgttg 26
<210> 586
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.019.2)
<400> 586
atctcgttga tatcctcaag gtcacc 26
<210> 587
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.020)
<400> 587
tcataccttc tgcttgatga tcatct 26
<210> 588
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.020.2)
<400> 588
tcattttttc tcataccttc tgcttg 26
<210> 589
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.021)
<400> 589
ttttctcata ccttctgctt gatgat 26
<210> 590
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.022)
<400> 590
ttttatcatt ttttctcata ccttct 26
<210> 591
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.25.023)
<400> 591
ccaactttta tcattttttc tcatac 26
<210> 592
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.001)
<400> 592
atattttggg tttttgcaaa 20
<210> 593
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.002)
<400> 593
aaaatatttt gggtttttgc 20
<210> 594
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.003)
<400> 594
gagctaaaat attttgggtt 20
<210> 595
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.004)
<400> 595
agtaggagct aaaatatttt 20
<210> 596
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.005)
<400> 596
gtctgagtag gagctaaaat 20
<210> 597
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.006)
<400> 597
taacagtctg agtaggagct 20
<210> 598
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.007)
<400> 598
cagagtaaca gtctgagtag 20
<210> 599
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.008)
<400> 599
cacaggttgt gtcaccagag 20
<210> 600
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.009)
<400> 600
agtttcctta gtaaccacag 20
<210> 601
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.010)
<400> 601
tagtttggag atggcagttt 20
<210> 602
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.011)
<400> 602
ggaagatggc atttctagtt 20
<210> 603
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.012)
<400> 603
tacctccaac atcaaggaag 20
<210> 604
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.013)
<400> 604
atctgccaga gcaggtacct 20
<210> 605
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.014)
<400> 605
ccaagcccgg ttgaaatctg 20
<210> 606
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.015)
<400> 606
gtcggtaagt tctgtccaag 20
<210> 607
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.016)
<400> 607
aagcagagaa agccagtcgg 20
<210> 608
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.017)
<400> 608
ttttataact tgatcaagca 20
<210> 609
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.018)
<400> 609
catcaccctc tgtgatttta 20
<210> 610
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.019)
<400> 610
ctcaaggtca cccaccatca 20
<210> 611
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.020)
<400> 611
catctcgttg atatcctcaa 20
<210> 612
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.021)
<400> 612
cttctgcttg atgatcatct 20
<210> 613
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.022)
<400> 613
cataccttct gcttgatgat 20
<210> 614
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.023)
<400> 614
tttctcatac cttctgcttg 20
<210> 615
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.024)
<400> 615
cattttttct cataccttct 20
<210> 616
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.025)
<400> 616
tttatcattt tttctcatac 20
<210> 617
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon51.20.026)
<400> 617
caacttttat cattttttct 20
<210> 618
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.001)
<400> 618
ctgtaagaac aaatatccct tagta 25
<210> 619
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.002)
<400> 619
tgcctgtaag aacaaatatc cctta 25
<210> 620
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.002.2)
<400> 620
gttgcctgta agaacaaata tccct 25
<210> 621
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.003)
<400> 621
attgttgcct gtaagaacaa atatc 25
<210> 622
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.003.2)
<400> 622
gcattgttgc ctgtaagaac aaata 25
<210> 623
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.004)
<400> 623
cctgcattgt tgcctgtaag aacaa 25
<210> 624
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.004.2)
<400> 624
atcctgcatt gttgcctgta agaac 25
<210> 625
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.005)
<400> 625
caaatcctgc attgttgcct gtaag 25
<210> 626
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.005.2)
<400> 626
tccaaatcct gcattgttgc ctgta 25
<210> 627
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.006)
<400> 627
tgttccaaat cctgcattgt tgcct 25
<210> 628
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.006.2)
<400> 628
tctgttccaa atcctgcatt gttgc 25
<210> 629
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.007)
<400> 629
aactggggac gcctctgttc caaat 25
<210> 630
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.007.2)
<400> 630
gcctctgttc caaatcctgc attgt 25
<210> 631
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.008)
<400> 631
cagcggtaat gagttcttcc aactg 25
<210> 632
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.008.2)
<400> 632
cttccaactg gggacgcctc tgttc 25
<210> 633
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.009)
<400> 633
cttgtttttc aaattttggg cagcg 25
<210> 634
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.010)
<400> 634
ctagcctctt gattgctggt cttgt 25
<210> 635
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.010.2)
<400> 635
ttttcaaatt ttgggcagcg gtaat 25
<210> 636
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.011)
<400> 636
ttcgatccgt aatgattgtt ctagc 25
<210> 637
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.011.2)
<400> 637
gattgctggt cttgtttttc aaatt 25
<210> 638
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.012)
<400> 638
cttacttcga tccgtaatga ttgtt 25
<210> 639
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.012.2)
<400> 639
ttgttctagc ctcttgattg ctggt 25
<210> 640
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.013)
<400> 640
aaaaacttac ttcgatccgt aatga 25
<210> 641
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.014)
<400> 641
tgttaaaaaa cttacttcga tccgt 25
<210> 642
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.015)
<400> 642
atgcttgtta aaaaacttac ttcga 25
<210> 643
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.25.016)
<400> 643
gtcccatgct tgttaaaaaa cttac 25
<210> 644
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.20.001)
<400> 644
agaacaaata tcccttagta 20
<210> 645
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.20.002)
<400> 645
gtaagaacaa atatccctta 20
<210> 646
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.20.003)
<400> 646
tgcctgtaag aacaaatatc 20
<210> 647
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.20.004)
<400> 647
attgttgcct gtaagaacaa 20
<210> 648
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.20.005)
<400> 648
cctgcattgt tgcctgtaag 20
<210> 649
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.20.006)
<400> 649
caaatcctgc attgttgcct 20
<210> 650
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.20.007)
<400> 650
gcctctgttc caaatcctgc 20
<210> 651
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.20.008)
<400> 651
cttccaactg gggacgcctc 20
<210> 652
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.20.009)
<400> 652
cagcggtaat gagttcttcc 20
<210> 653
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.20.010)
<400> 653
ttttcaaatt ttgggcagcg 20
<210> 654
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.20.011)
<400> 654
gattgctggt cttgtttttc 20
<210> 655
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.20.012)
<400> 655
ttgttctagc ctcttgattg 20
<210> 656
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.20.013)
<400> 656
ttcgatccgt aatgattgtt 20
<210> 657
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.20.014)
<400> 657
cttacttcga tccgtaatga 20
<210> 658
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.20.015)
<400> 658
aaaaacttac ttcgatccgt 20
<210> 659
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.20.016)
<400> 659
tgttaaaaaa cttacttcga 20
<210> 660
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.20.017)
<400> 660
atgcttgtta aaaaacttac 20
<210> 661
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon52.20.018)
<400> 661
gtcccatgct tgttaaaaaa 20
<210> 662
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.001)
<400> 662
ctagaataaa aggaaaaata aatat 25
<210> 663
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.002)
<400> 663
aactagaata aaaggaaaaa taaat 25
<210> 664
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.002.2)
<400> 664
ttcaactaga ataaaaggaa aaata 25
<210> 665
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.003)
<400> 665
ctttcaacta gaataaaagg aaaaa 25
<210> 666
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.003.2)
<400> 666
attctttcaa ctagaataaa aggaa 25
<210> 667
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.004)
<400> 667
gaattctttc aactagaata aaagg 25
<210> 668
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.004.2)
<400> 668
tctgaattct ttcaactaga ataaa 25
<210> 669
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.005)
<400> 669
attctgaatt ctttcaacta gaata 25
<210> 670
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.005.2)
<400> 670
ctgattctga attctttcaa ctaga 25
<210> 671
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.006)
<400> 671
cactgattct gaattctttc aacta 25
<210> 672
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.006.2)
<400> 672
tcccactgat tctgaattct ttcaa 25
<210> 673
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.007)
<400> 673
catcccactg attctgaatt ctttc 25
<210> 674
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.008)
<400> 674
tacttcatcc cactgattct gaatt 25
<210> 675
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.009)
<400> 675
cggttctgaa ggtgttcttg tact 24
<210> 676
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.009.2)
<400> 676
ctgttgcctc cggttctgaa ggtgt 25
<210> 677
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.010)
<400> 677
tttcattcaa ctgttgcctc cggtt 25
<210> 678
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.010.2)
<400> 678
taacatttca ttcaactgtt gcctc 25
<210> 679
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.011)
<400> 679
ttgtgttgaa tcctttaaca tttca 25
<210> 680
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.012)
<400> 680
tcttccttag cttccagcca ttgtg 25
<210> 681
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.012.2)
<400> 681
cttagcttcc agccattgtg ttgaa 25
<210> 682
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.013)
<400> 682
gtcctaagac ctgctcagct tcttc 25
<210> 683
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.013.2)
<400> 683
ctgctcagct tcttccttag cttcc 25
<210> 684
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.014)
<400> 684
ctcaagcttg gctctggcct gtcct 25
<210> 685
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.014.2)
<400> 685
ggcctgtcct aagacctgct cagct 25
<210> 686
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.015)
<400> 686
tagggaccct ccttccatga ctcaa 25
<210> 687
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.016)
<400> 687
tttggattgc atctactgta taggg 25
<210> 688
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.016.2)
<400> 688
accctccttc catgactcaa gcttg 25
<210> 689
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.017)
<400> 689
cttggtttct gtgattttct tttgg 25
<210> 690
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.017.2)
<400> 690
atctactgta tagggaccct ccttc 25
<210> 691
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.018)
<400> 691
ctaaccttgg tttctgtgat tttct 25
<210> 692
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.018.2)
<400> 692
tttcttttgg attgcatcta ctgta 25
<210> 693
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.019)
<400> 693
tgatactaac cttggtttct gtgat 25
<210> 694
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.020)
<400> 694
atctttgata ctaaccttgg tttct 25
<210> 695
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.021)
<400> 695
aaggtatctt tgatactaac cttgg 25
<210> 696
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.25.022)
<400> 696
ttaaaaaggt atctttgata ctaac 25
<210> 697
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.001)
<400> 697
ataaaaggaa aaataaatat 20
<210> 698
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.002)
<400> 698
gaataaaagg aaaaataaat 20
<210> 699
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.003)
<400> 699
aactagaata aaaggaaaaa 20
<210> 700
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.004)
<400> 700
ctttcaacta gaataaaagg 20
<210> 701
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.005)
<400> 701
gaattctttc aactagaata 20
<210> 702
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.006)
<400> 702
attctgaatt ctttcaacta 20
<210> 703
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.007)
<400> 703
tacttcatcc cactgattct 20
<210> 704
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.008)
<400> 704
ctgaaggtgt tcttgtact 19
<210> 705
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.009)
<400> 705
ctgttgcctc cggttctgaa 20
<210> 706
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.010)
<400> 706
taacatttca ttcaactgtt 20
<210> 707
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.011)
<400> 707
ttgtgttgaa tcctttaaca 20
<210> 708
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.012)
<400> 708
cttagcttcc agccattgtg 20
<210> 709
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.013)
<400> 709
ctgctcagct tcttccttag 20
<210> 710
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.014)
<400> 710
ggcctgtcct aagacctgct 20
<210> 711
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.015)
<400> 711
ctcaagcttg gctctggcct 20
<210> 712
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.016)
<400> 712
accctccttc catgactcaa 20
<210> 713
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.017)
<400> 713
atctactgta tagggaccct 20
<210> 714
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.018)
<400> 714
tttcttttgg attgcatcta 20
<210> 715
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.019)
<400> 715
cttggtttct gtgattttct 20
<210> 716
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.020)
<400> 716
ctaaccttgg tttctgtgat 20
<210> 717
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.021)
<400> 717
tgatactaac cttggtttct 20
<210> 718
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.022)
<400> 718
atctttgata ctaaccttgg 20
<210> 719
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.023)
<400> 719
aaggtatctt tgatactaac 20
<210> 720
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon53.20.024)
<400> 720
ttaaaaaggt atctttgata 20
<210> 721
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.001)
<400> 721
ctatagattt ttatgagaaa gaga 24
<210> 722
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.002)
<400> 722
aactgctata gatttttatg agaaa 25
<210> 723
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.003)
<400> 723
tggccaactg ctatagattt ttatg 25
<210> 724
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.004)
<400> 724
gtctttggcc aactgctata gattt 25
<210> 725
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.005)
<400> 725
cggaggtctt tggccaactg ctata 25
<210> 726
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.006)
<400> 726
actggcggag gtctttggcc aactg 25
<210> 727
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.007)
<400> 727
tttgtctgcc actggcggag gtctt 25
<210> 728
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.008)
<400> 728
agtcatttgc cacatctaca tttgt 25
<210> 729
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.008.2)
<400> 729
tttgccacat ctacatttgt ctgcc 25
<210> 730
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.009)
<400> 730
ccggagaagt ttcagggcca agtca 25
<210> 731
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.010)
<400> 731
gtatcatctg cagaataatc ccgga 25
<210> 732
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.010.2)
<400> 732
taatcccgga gaagtttcag ggcca 25
<210> 733
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.011)
<400> 733
ttatcatgtg gacttttctg gtatc 25
<210> 734
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.012)
<400> 734
agaggcattg atattctctg ttatc 25
<210> 735
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.012.2)
<400> 735
atgtggactt ttctggtatc atctg 25
<210> 736
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.013)
<400> 736
cttttatgaa tgcttctcca agagg 25
<210> 737
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.013.2)
<400> 737
atattctctg ttatcatgtg gactt 25
<210> 738
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.014)
<400> 738
catacctttt atgaatgctt ctcca 25
<210> 739
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.014.2)
<400> 739
ctccaagagg cattgatatt ctctg 25
<210> 740
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.015)
<400> 740
taattcatac cttttatgaa tgctt 25
<210> 741
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.016)
<400> 741
taatgtaatt catacctttt atgaa 25
<210> 742
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.017)
<400> 742
agaaataatg taattcatac ctttt 25
<210> 743
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.25.018)
<400> 743
gttttagaaa taatgtaatt catac 25
<210> 744
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.20.001)
<400> 744
gatttttatg agaaagaga 19
<210> 745
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.20.002)
<400> 745
ctatagattt ttatgagaaa 20
<210> 746
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.20.003)
<400> 746
aactgctata gatttttatg 20
<210> 747
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.20.004)
<400> 747
tggccaactg ctatagattt 20
<210> 748
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.20.005)
<400> 748
gtctttggcc aactgctata 20
<210> 749
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.20.006)
<400> 749
cggaggtctt tggccaactg 20
<210> 750
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.20.007)
<400> 750
tttgtctgcc actggcggag 20
<210> 751
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.20.008)
<400> 751
tttgccacat ctacatttgt 20
<210> 752
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.20.009)
<400> 752
ttcagggcca agtcatttgc 20
<210> 753
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.20.010)
<400> 753
taatcccgga gaagtttcag 20
<210> 754
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.20.011)
<400> 754
gtatcatctg cagaataatc 20
<210> 755
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.20.012)
<400> 755
atgtggactt ttctggtatc 20
<210> 756
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.20.013)
<400> 756
atattctctg ttatcatgtg 20
<210> 757
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.20.014)
<400> 757
ctccaagagg cattgatatt 20
<210> 758
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.20.015)
<400> 758
cttttatgaa tgcttctcca 20
<210> 759
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.20.016)
<400> 759
catacctttt atgaatgctt 20
<210> 760
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.20.017)
<400> 760
taattcatac cttttatgaa 20
<210> 761
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.20.018)
<400> 761
taatgtaatt catacctttt 20
<210> 762
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.20.019)
<400> 762
agaaataatg taattcatac 20
<210> 763
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon54.20.020)
<400> 763
gttttagaaa taatgtaatt 20
<210> 764
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.001)
<400> 764
ctgcaaagga ccaaatgttc agatg 25
<210> 765
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.002)
<400> 765
tcaccctgca aaggaccaaa tgttc 25
<210> 766
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.003)
<400> 766
ctcactcacc ctgcaaagga ccaaa 25
<210> 767
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.004)
<400> 767
tctcgctcac tcaccctgca aagga 25
<210> 768
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.005)
<400> 768
cagcctctcg ctcactcacc ctgca 25
<210> 769
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.006)
<400> 769
caaagcagcc tctcgctcac tcacc 25
<210> 770
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.007)
<400> 770
tcttccaaag cagcctctcg ctcac 25
<210> 771
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.007.2)
<400> 771
tctatgagtt tcttccaaag cagcc 25
<210> 772
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.008)
<400> 772
gttgcagtaa tctatgagtt tcttc 25
<210> 773
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.008.2)
<400> 773
gaactgttgc agtaatctat gagtt 25
<210> 774
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.009)
<400> 774
ttccaggtcc agggggaact gttgc 25
<210> 775
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.010)
<400> 775
gtaagccagg caagaaactt ttcca 25
<210> 776
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.010.2)
<400> 776
ccaggcaaga aacttttcca ggtcc 25
<210> 777
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.011)
<400> 777
tggcagttgt ttcagcttct gtaag 25
<210> 778
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.011.2)
<400> 778
ttcagcttct gtaagccagg caaga 25
<210> 779
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.012)
<400> 779
ggtagcatcc tgtaggacat tggca 25
<210> 780
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.012.2)
<400> 780
gacattggca gttgtttcag cttct 25
<210> 781
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.013)
<400> 781
tctaggagcc tttccttacg ggtag 25
<210> 782
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.014)
<400> 782
cttttactcc cttggagtct tctag 25
<210> 783
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.014.2)
<400> 783
gagcctttcc ttacgggtag catcc 25
<210> 784
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.015)
<400> 784
ttgccattgt ttcatcagct ctttt 25
<210> 785
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.015.2)
<400> 785
cttggagtct tctaggagcc tttcc 25
<210> 786
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.016)
<400> 786
cttacttgcc attgtttcat cagct 25
<210> 787
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.016.2)
<400> 787
cagctctttt actcccttgg agtct 25
<210> 788
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.017)
<400> 788
cctgacttac ttgccattgt ttcat 25
<210> 789
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.018)
<400> 789
aaatgcctga cttacttgcc attgt 25
<210> 790
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.019)
<400> 790
agcggaaatg cctgacttac ttgcc 25
<210> 791
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.25.020)
<400> 791
gctaaagcgg aaatgcctga cttac 25
<210> 792
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.001)
<400> 792
aaggaccaaa tgttcagatg 20
<210> 793
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.002)
<400> 793
ctgcaaagga ccaaatgttc 20
<210> 794
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.003)
<400> 794
tcaccctgca aaggaccaaa 20
<210> 795
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.004)
<400> 795
ctcactcacc ctgcaaagga 20
<210> 796
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.005)
<400> 796
tctcgctcac tcaccctgca 20
<210> 797
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.006)
<400> 797
cagcctctcg ctcactcacc 20
<210> 798
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.007)
<400> 798
caaagcagcc tctcgctcac 20
<210> 799
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.008)
<400> 799
tctatgagtt tcttccaaag 20
<210> 800
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.009)
<400> 800
gaactgttgc agtaatctat 20
<210> 801
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.010)
<400> 801
ttccaggtcc agggggaact 20
<210> 802
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.011)
<400> 802
ccaggcaaga aacttttcca 20
<210> 803
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.012)
<400> 803
ttcagcttct gtaagccagg 20
<210> 804
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.013)
<400> 804
gacattggca gttgtttcag 20
<210> 805
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.014)
<400> 805
ggtagcatcc tgtaggacat 20
<210> 806
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.015)
<400> 806
gagcctttcc ttacgggtag 20
<210> 807
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.016)
<400> 807
cttggagtct tctaggagcc 20
<210> 808
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.017)
<400> 808
cagctctttt actcccttgg 20
<210> 809
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.018)
<400> 809
ttgccattgt ttcatcagct 20
<210> 810
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.019)
<400> 810
cttacttgcc attgtttcat 20
<210> 811
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.020)
<400> 811
cctgacttac ttgccattgt 20
<210> 812
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.021)
<400> 812
aaatgcctga cttacttgcc 20
<210> 813
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.022)
<400> 813
agcggaaatg cctgacttac 20
<210> 814
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (Hu.DMD.Exon55.20.023)
<400> 814
gctaaagcgg aaatgcctga 20
<210> 815
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H50A(+02+30)-AVI-5656)
<400> 815
ccactcagag ctcagatctt ctaacttcc 29
<210> 816
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H50D(+07-18)-AVI-5915)
<400> 816
gggatccagt atacttacag gctcc 25
<210> 817
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H50A(+07+33))
<400> 817
cttccactca gagctcagat cttctaa 27
<210> 818
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H51A(+61+90)-AVI-4657)
<400> 818
acatcaagga agatggcatt tctagtttgg 30
<210> 819
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H51A(+66+95)-AVI-4658)
<400> 819
ctccaacatc aaggaagatg gcatttctag 30
<210> 820
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H51A(+111+134))
<400> 820
ttctgtccaa gcccggttga aatc 24
<210> 821
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H51A(+175+195))
<400> 821
cacccaccat caccctcygt g 21
<210> 822
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H51A(+199+220))
<400> 822
atcatctcgt tgatatcctc aa 22
<210> 823
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H51A(+66+90))
<400> 823
acatcaagga agatggcatt tctag 25
<210> 824
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H51A(-01+25))
<400> 824
accagagtaa cagtctgagt aggagc 26
<210> 825
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (h51AON1)
<400> 825
tcaaggaaga tggcatttct 20
<210> 826
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (h51AON2)
<400> 826
cctctgtgat tttataactt gat 23
<210> 827
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H51D(+08-17))
<400> 827
atcatttttt ctcatacctt ctgct 25
<210> 828
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H51D(+16-07))
<400> 828
ctcatacctt ctgcttgatg atc 23
<210> 829
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (hAON#23)
<400> 829
tggcatttct agtttgg 17
<210> 830
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (hAON#24)
<400> 830
ccagagcagg tacctccaac atc 23
<210> 831
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (h44AON1)
<400> 831
cgccgccatt tctcaacag 19
<210> 832
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H45A(+71+90))
<400> 832
tgtttttgag gattgctgaa 20
<210> 833
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (h45AON1)
<400> 833
gctgaattat ttcttcccc 19
<210> 834
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (h45AON5)
<400> 834
gcccaatgcc atcctgg 17
<210> 835
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H45A(-06+20))
<400> 835
ccaatgccat cctggagttc ctgtaa 26
<210> 836
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(+39+69))
<400> 836
cattcaactg ttgcctccgg ttctgaaggt g 31
<210> 837
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (h53AON1)
<400> 837
ctgttgcctc cggttctg 18
<210> 838
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (H53A(-12+10))
<400> 838
attctttcaa ctagaataaa ag 22
<210> 839
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (huEx45.30.66)
<400> 839
gccatcctgg agttcctgta agataccaaa 30
<210> 840
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (huEx45.30.71)
<400> 840
ccaatgccat cctggagttc ctgtaagata 30
<210> 841
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (huEx45.30.79)
<400> 841
gccgctgccc aatgccatcc tggagttcct 30
<210> 842
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (huEx45.30.83)
<400> 842
gtttgccgct gcccaatgcc atcctggagt 30
<210> 843
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (huEx45.30.88)
<400> 843
caacagtttg ccgctgccca atgccatcct 30
<210> 844
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (huEx45.30.92)
<400> 844
ctgacaacag tttgccgctg cccaatgcca 30
<210> 845
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (huEx45.30.96)
<400> 845
tgttctgaca acagtttgcc gctgcccaat 30
<210> 846
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (huEx45.30.99)
<400> 846
caatgttctg acaacagttt gccgctgccc 30
<210> 847
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (huEx45.30.103)
<400> 847
cattcaatgt tctgacaaca gtttgccgct 30
<210> 848
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (huEx45.30.120)
<400> 848
tatttcttcc ccagttgcat tcaatgttct 30
<210> 849
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (huEx45.30.127)
<400> 849
gctgaattat ttcttcccca gttgcattca 30
<210> 850
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (huEx45.30.132)
<400> 850
ggattgctga attatttctt ccccagttgc 30
<210> 851
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (huEx45.30.137)
<400> 851
tttgaggatt gctgaattat ttcttcccca 30
<210> 852
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (huEx53.30.84)
<400> 852
gtacttcatc ccactgattc tgaattcttt 30
<210> 853
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (huEx53.30.88)
<400> 853
tcttgtactt catcccactg attctgaatt 30
<210> 854
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (huEx53.30.91)
<400> 854
tgttcttgta cttcatccca ctgattctga 30
<210> 855
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (huEx53.30.103)
<400> 855
cggttctgaa ggtgttcttg tacttcatcc 30
<210> 856
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (huEx53.30.106)
<400> 856
ctccggttct gaaggtgttc ttgtacttca 30
<210> 857
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (huEx53.30.109)
<400> 857
tgcctccggt tctgaaggtg ttcttgtact 30
<210> 858
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (huEx53.30.112)
<400> 858
tgttgcctcc ggttctgaag gtgttcttgt 30
<210> 859
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (huEx53.30.115)
<400> 859
aactgttgcc tccggttctg aaggtgttct 30
<210> 860
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (huEx53.30.118)
<400> 860
ttcaactgtt gcctccggtt ctgaaggtgt 30
<210> 861
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic (rTAT)
<400> 861
Arg Arg Arg Gln Arg Arg Lys Lys Arg Cys
1 5 10
<210> 862
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic (R9F2)
<400> 862
Arg Arg Arg Arg Arg Arg Arg Arg Arg Phe Phe Cys
1 5 10
<210> 863
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic ((RRAhx)4B)
<220>
<221> misc_feature
<222> (3)..(3)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (6)..(6)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (9)..(9)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (12)..(12)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (13)..(13)
<223> Xaa is beta-alanine
<400> 863
Arg Arg Xaa Arg Arg Xaa Arg Arg Xaa Arg Arg Xaa Xaa
1 5 10
<210> 864
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic ((RAhxR)4AhxB; (P007) )
<220>
<221> misc_feature
<222> (2)..(2)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (5)..(5)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (8)..(8)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (11)..(11)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (13)..(13)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (14)..(14)
<223> Xaa is beta-alanine
<400> 864
Arg Xaa Arg Arg Xaa Arg Arg Xaa Arg Arg Xaa Arg Xaa Xaa
1 5 10
<210> 865
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic ((AhxRR)4AhxB)
<220>
<221> misc_feature
<222> (1)..(1)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (4)..(4)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (7)..(7)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (10)..(10)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (13)..(13)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (14)..(14)
<223> Xaa is beta-alanine
<400> 865
Xaa Arg Arg Xaa Arg Arg Xaa Arg Arg Xaa Arg Arg Xaa Xaa
1 5 10
<210> 866
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic ((RAhx)6B)
<220>
<221> misc_feature
<222> (2)..(2)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (4)..(4)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (6)..(6)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (8)..(8)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (10)..(10)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (12)..(12)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (13)..(13)
<223> Xaa is beta-alanine
<400> 866
Arg Xaa Arg Xaa Arg Xaa Arg Xaa Arg Xaa Arg Xaa Xaa
1 5 10
<210> 867
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic ((RAhx)8B)
<220>
<221> misc_feature
<222> (2)..(2)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (4)..(4)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (6)..(6)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (8)..(8)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (10)..(10)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (12)..(12)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (14)..(14)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (15)..(15)
<223> Xaa is beta-alanine
<400> 867
Arg Xaa Arg Xaa Arg Xaa Arg Xaa Arg Xaa Arg Xaa Arg Xaa Xaa
1 5 10 15
<210> 868
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic ((RAhxR)5AhxB)
<220>
<221> misc_feature
<222> (2)..(2)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (5)..(5)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (8)..(8)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (11)..(11)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (14)..(14)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (16)..(16)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (17)..(17)
<223> Xaa is beta-alanine
<400> 868
Arg Xaa Arg Arg Xaa Arg Arg Xaa Arg Arg Xaa Arg Arg Xaa Arg Xaa
1 5 10 15
Xaa
<210> 869
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic ((RAhxRRBR)2AhxB; (CPO6062))
<220>
<221> misc_feature
<222> (2)..(2)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (5)..(5)
<223> Xaa is beta-alanine
<220>
<221> misc_feature
<222> (8)..(8)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (11)..(11)
<223> Xaa is beta-alanine
<220>
<221> misc_feature
<222> (13)..(13)
<223> Xaa is 6-aminohexanoic acid (Ahx)
<220>
<221> misc_feature
<222> (14)..(14)
<223> Xaa is beta-alanine
<400> 869
Arg Xaa Arg Arg Xaa Arg Arg Xaa Arg Arg Xaa Arg Xaa Xaa
1 5 10
<210> 870
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic (MSP)
<400> 870
Ala Ser Ser Leu Asn Ile Ala
1 5
<210> 871
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic (MSP-PMO)
<220>
<221> misc_feature
<222> (8)..(9)
<223> Xaa can be any naturally occurring amino acid
<400> 871
Ala Ser Ser Leu Asn Ile Ala Xaa Xaa
1 5
<210> 872
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic (CP06062-MSP-PMO)
<220>
<221> misc_feature
<222> (2)..(2)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (5)..(5)
<223> Xaa is beta-alanine
<220>
<221> misc_feature
<222> (8)..(8)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (11)..(11)
<223> Xaa is beta-alanine
<220>
<221> misc_feature
<222> (13)..(13)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (14)..(14)
<223> Xaa is beta-alanine
<220>
<221> misc_feature
<222> (22)..(22)
<223> Xaa can be any naturally occurring amino acid
<400> 872
Arg Xaa Arg Arg Xaa Arg Arg Xaa Arg Arg Xaa Arg Xaa Xaa Ala Ser
1 5 10 15
Ser Leu Asn Ile Ala Xaa
20
<210> 873
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic (MSP-CP06062-PMO)
<220>
<221> misc_feature
<222> (8)..(8)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (10)..(10)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (13)..(13)
<223> Xaa is beta-alanine
<220>
<221> misc_feature
<222> (16)..(16)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (19)..(19)
<223> Xaa is beta-alanine
<220>
<221> misc_feature
<222> (21)..(21)
<223> Xaa is beta-alanine
<400> 873
Ala Ser Ser Leu Asn Ile Ala Xaa Arg Xaa Arg Arg Xaa Arg Arg Xaa
1 5 10 15
Arg Arg Xaa Arg Xaa
20
<210> 874
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic (CP06062-PMO)
<220>
<221> misc_feature
<222> (2)..(2)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (5)..(5)
<223> Xaa is beta-alanine
<220>
<221> misc_feature
<222> (8)..(8)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (11)..(11)
<223> Xaa is beta-alanine
<220>
<221> misc_feature
<222> (13)..(13)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (14)..(14)
<223> Xaa is beta-alanine
<400> 874
Arg Xaa Arg Arg Xaa Arg Arg Xaa Arg Arg Xaa Arg Xaa Xaa
1 5 10
<210> 875
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic (PMO)
<400> 875
ggccaaacct cggcttacct gaaat 25
<210> 876
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic (rTAT)
<400> 876
Arg Arg Arg Gln Arg Arg Lys Lys Arg
1 5
<210> 877
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic (Tat)
<400> 877
Arg Lys Lys Arg Arg Gln Arg Arg Arg
1 5
<210> 878
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic (R9F2)
<400> 878
Arg Arg Arg Arg Arg Arg Arg Arg Arg Phe Phe
1 5 10
<210> 879
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic (R5F2R4)
<400> 879
Arg Arg Arg Arg Arg Phe Phe Arg Arg Arg Arg
1 5 10
<210> 880
<211> 4
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic (R4)
<400> 880
Arg Arg Arg Arg
1
<210> 881
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic (R5)
<400> 881
Arg Arg Arg Arg Arg
1 5
<210> 882
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic (R6)
<400> 882
Arg Arg Arg Arg Arg Arg
1 5
<210> 883
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic (R7)
<400> 883
Arg Arg Arg Arg Arg Arg Arg
1 5
<210> 884
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic (R8)
<400> 884
Arg Arg Arg Arg Arg Arg Arg Arg
1 5
<210> 885
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic (R9)
<400> 885
Arg Arg Arg Arg Arg Arg Arg Arg Arg
1 5
<210> 886
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic ((RX)8)
<220>
<221> misc_feature
<222> (2)..(2)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (4)..(4)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (6)..(6)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (8)..(8)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (10)..(10)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (12)..(12)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (14)..(14)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (16)..(16)
<223> Xaa can be any naturally occurring amino acid
<400> 886
Arg Xaa Arg Xaa Arg Xaa Arg Xaa Arg Xaa Arg Xaa Arg Xaa Arg Xaa
1 5 10 15
<210> 887
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic ((RAhxR)4; (P007) )
<220>
<221> misc_feature
<222> (2)..(2)
<223> Xaa is 6-aminohexanoic acid
<220>
<221> misc_feature
<222> (5)..(5)
<223> Xaa is 6-aminohexanoic acid
<220>
<221> misc_feature
<222> (8)..(8)
<223> Xaa is 6-aminohexanoic acid
<220>
<221> misc_feature
<222> (11)..(11)
<223> Xaa is 6-aminohexanoic acid
<400> 887
Arg Xaa Arg Arg Xaa Arg Arg Xaa Arg Arg Xaa Arg
1 5 10
<210> 888
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic ((RAhxR)5; (CP04057))
<220>
<221> misc_feature
<222> (2)..(2)
<223> Xaa is 6-aminohexanoic acid
<220>
<221> misc_feature
<222> (5)..(5)
<223> Xaa is 6-aminohexanoic acid
<220>
<221> misc_feature
<222> (8)..(8)
<223> Xaa is 6-aminohexanoic acid
<220>
<221> misc_feature
<222> (11)..(11)
<223> Xaa is 6-aminohexanoic acid
<220>
<221> misc_feature
<222> (14)..(14)
<223> Xaa is 6-aminohexanoic acid
<400> 888
Arg Xaa Arg Arg Xaa Arg Arg Xaa Arg Arg Xaa Arg Arg Xaa Arg
1 5 10 15
<210> 889
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic ((RAhxRRBR)2; (CP06062))
<220>
<221> misc_feature
<222> (2)..(2)
<223> Xaa is 6-aminohexanoic acid
<220>
<221> misc_feature
<222> (5)..(5)
<223> Xaa is beta-alanine
<220>
<221> misc_feature
<222> (8)..(8)
<223> Xaa is 6-aminohexanoic acid
<220>
<221> misc_feature
<222> (11)..(11)
<223> Xaa is beta-alanine
<400> 889
Arg Xaa Arg Arg Xaa Arg Arg Xaa Arg Arg Xaa Arg
1 5 10
<210> 890
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic ((RAR)4F2)
<400> 890
Arg Ala Arg Arg Ala Arg Arg Ala Arg Arg Ala Arg Phe Phe
1 5 10
<210> 891
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic ((RGR)4F2))
<400> 891
Arg Gly Arg Arg Gly Arg Arg Gly Arg Arg Gly Arg Phe Phe
1 5 10
Claims (26)
- 뒤시엔느 근육 이영양증의 치료를 필요로 하는 환자에게 약 30 mg/kg의 용량의 에테플러센을 주 1회 투여하는 것을 포함하는, 상기 환자에서 뒤시엔느 근육 이영양증을 치료하는 방법.
- 제1항에 있어서, 에테플러센이 단일 용량으로 투여되는 것인 방법.
- 제1항에 있어서, 에테플러센이 정맥내로 투여되는 것인 방법.
- 제1항에 있어서, 환자가 디스트로핀 유전자의 엑손 51의 스키핑에 의해 교정될 수 있는 아웃-오브-프레임(out-of-frame) 결실(들)을 갖는 환자인 방법.
- 제1항에 있어서, 환자가 소아 환자인 방법.
- 제1항에 있어서, 환자가 제1 용량의 에테플러센 투여 전에 적어도 24주 동안 경구 코르티코스테로이드를 투여받는 것인 방법.
- 뒤시엔느 근육 이영양증의 치료를 필요로 하는 환자에게 단일 용량의 약 30 mg/kg의 에테플러센을 주 1회 정맥내로 투여하는 것을 포함하며, 여기서 상기 환자는 디스트로핀 유전자의 엑손 51의 스키핑에 의해 교정될 수 있는 아웃-오브-프레임 결실(들)을 갖는 환자인, 상기 환자에서 뒤시엔느 근육 이영양증을 치료하는 방법.
- 뒤시엔느 근육 이영양증의 치료를 필요로 하는 환자에게 약 50 mg/kg의 용량의 에테플러센을 주 1회 투여하는 것을 포함하는, 상기 환자에서 뒤시엔느 근육 이영양증을 치료하는 방법.
- 제8항에 있어서, 에테플러센이 단일 용량으로 투여되는 것인 방법.
- 제8항에 있어서, 에테플러센이 정맥내로 투여되는 것인 방법.
- 제8항에 있어서, 환자가 디스트로핀 유전자의 엑손 51의 스키핑에 의해 교정될 수 있는 아웃-오브-프레임 결실(들)을 갖는 환자인 방법.
- 제8항에 있어서, 환자가 소아 환자인 방법.
- 제8항에 있어서, 환자가 제1 용량의 에테플러센 투여 전에 적어도 24주 동안 경구 코르티코스테로이드를 투여받는 것인 방법.
- 뒤시엔느 근육 이영양증의 치료를 필요로 하는 환자에게 단일 용량의 약 50 mg/kg의 에테플러센을 주 1회 정맥내로 투여하는 것을 포함하며, 여기서 상기 환자는 디스트로핀 유전자의 엑손 51의 스키핑에 의해 교정될 수 있는 아웃-오브-프레임 결실(들)을 갖는 환자인, 상기 환자에서 뒤시엔느 근육 이영양증을 치료하는 방법.
- 뒤시엔느 근육 이영양증을 갖는 환자에게 약 30 mg/kg의 용량의 에테플러센을 주 1회 투여하는 것을 포함하는, 상기 환자에서 디스트로핀 제조를 증가시키는 방법.
- 제15항에 있어서, 에테플러센이 단일 용량으로 투여되는 것인 방법.
- 제15항에 있어서, 에테플러센이 정맥내로 투여되는 것인 방법.
- 제15항에 있어서, 환자가 디스트로핀 유전자의 엑손 51의 스키핑에 의해 교정될 수 있는 아웃-오브-프레임 결실(들)을 갖는 환자인 방법.
- 제15항에 있어서, 환자가 소아 환자인 방법.
- 제15항에 있어서, 환자가 제1 용량의 에테플러센 투여 전에 적어도 24주 동안 경구 코르티코스테로이드를 투여받는 것인 방법.
- 뒤시엔느 근육 이영양증을 갖는 환자에게 약 50 mg/kg의 용량의 에테플러센을 주 1회 투여하는 것을 포함하는, 상기 환자에서 디스트로핀 제조를 증가시키는 방법.
- 제21항에 있어서, 에테플러센이 단일 용량으로 투여되는 것인 방법.
- 제21항에 있어서, 에테플러센이 정맥내로 투여되는 것인 방법.
- 제21항에 있어서, 환자가 디스트로핀 유전자의 엑손 51의 스키핑에 의해 교정될 수 있는 아웃-오브-프레임 결실(들)을 갖는 환자인 방법.
- 제21항에 있어서, 환자가 소아 환자인 방법.
- 제21항에 있어서, 환자가 제1 용량의 에테플러센 투여 전에 적어도 24주 동안 경구 코르티코스테로이드를 투여받는 것인 방법.
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201361793463P | 2013-03-15 | 2013-03-15 | |
| US61/793,463 | 2013-03-15 | ||
| PCT/US2014/029610 WO2014144978A2 (en) | 2013-03-15 | 2014-03-14 | Improved compositions for treating muscular dystrophy |
Related Child Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020207034743A Division KR20200139271A (ko) | 2013-03-15 | 2014-03-14 | 근육 이영양증의 치료를 위한 개선된 조성물 |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20150133768A true KR20150133768A (ko) | 2015-11-30 |
Family
ID=50678303
Family Applications (2)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020207034743A KR20200139271A (ko) | 2013-03-15 | 2014-03-14 | 근육 이영양증의 치료를 위한 개선된 조성물 |
| KR1020157029546A KR20150133768A (ko) | 2013-03-15 | 2014-03-14 | 근육 이영양증의 치료를 위한 개선된 조성물 |
Family Applications Before (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020207034743A KR20200139271A (ko) | 2013-03-15 | 2014-03-14 | 근육 이영양증의 치료를 위한 개선된 조성물 |
Country Status (14)
| Country | Link |
|---|---|
| US (8) | US20140329762A1 (ko) |
| EP (2) | EP2968991A2 (ko) |
| JP (2) | JP2016516066A (ko) |
| KR (2) | KR20200139271A (ko) |
| CN (2) | CN105307723A (ko) |
| AU (3) | AU2014233456B2 (ko) |
| BR (1) | BR112015022998A2 (ko) |
| CA (1) | CA2906812A1 (ko) |
| EA (1) | EA201591792A1 (ko) |
| HK (1) | HK1220154A1 (ko) |
| IL (3) | IL241558B (ko) |
| MX (2) | MX2015013117A (ko) |
| NZ (2) | NZ631289A (ko) |
| WO (1) | WO2014144978A2 (ko) |
Families Citing this family (74)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| EP3808845A1 (en) | 2004-06-28 | 2021-04-21 | The University Of Western Australia | Antisense oligonucleotides for inducing exon skipping and methods of use thereof |
| EP1855694B1 (en) | 2005-02-09 | 2020-12-02 | Sarepta Therapeutics, Inc. | Antisense composition for treating muscle atrophy |
| US8871918B2 (en) | 2008-10-24 | 2014-10-28 | Sarepta Therapeutics, Inc. | Multiple exon skipping compositions for DMD |
| KR102581868B1 (ko) | 2009-11-12 | 2023-10-04 | 더 유니버시티 오브 웨스턴 오스트레일리아 | 안티센스 분자 및 이를 이용한 질환 치료방법 |
| TWI541024B (zh) | 2010-09-01 | 2016-07-11 | 日本新藥股份有限公司 | 反義核酸 |
| US20130085139A1 (en) | 2011-10-04 | 2013-04-04 | Royal Holloway And Bedford New College | Oligomers |
| WO2013163628A2 (en) | 2012-04-27 | 2013-10-31 | Duke University | Genetic correction of mutated genes |
| CA2906209A1 (en) | 2013-03-14 | 2014-09-25 | Sarepta Therapeutics, Inc. | Exon skipping compositions for treating muscular dystrophy targeting the annealing site h44a (-07+15) |
| JP2016516066A (ja) | 2013-03-15 | 2016-06-02 | サレプタ セラピューティクス, インコーポレイテッド | 筋ジストロフィを処置するための改善された組成物 |
| AU2015311708A1 (en) | 2014-09-07 | 2017-02-02 | Selecta Biosciences, Inc. | Methods and compositions for attenuating exon skipping anti-viral transfer vector immune responses |
| MA41795A (fr) | 2015-03-18 | 2018-01-23 | Sarepta Therapeutics Inc | Exclusion d'un exon induite par des composés antisens dans la myostatine |
| KR20240035901A (ko) | 2015-05-19 | 2024-03-18 | 사렙타 쎄러퓨틱스 인코퍼레이티드 | 펩티드 올리고뉴클레오티드 콘주게이트 |
| CN108026531B (zh) | 2015-09-15 | 2021-09-14 | 日本新药株式会社 | 反义核酸 |
| CN108738310A (zh) * | 2015-09-30 | 2018-11-02 | 萨勒普塔医疗公司 | 用于治疗肌营养不良的方法 |
| IL295755A (en) | 2015-10-09 | 2022-10-01 | Wave Life Sciences Ltd | Oligonucleotide preparations and methods thereof |
| EP3359668A4 (en) | 2015-10-09 | 2019-06-05 | Sarepta Therapeutics, Inc. | COMPOSITIONS AND METHODS OF TREATING DUCHENNE MUSCLE DYSTROPHY AND ASSOCIATED ILLNESSES THEREOF |
| EP4089175A1 (en) | 2015-10-13 | 2022-11-16 | Duke University | Genome engineering with type i crispr systems in eukaryotic cells |
| BR112018011133A2 (pt) * | 2015-11-30 | 2018-11-21 | Univ Duke | alvos terapêuticos para a correção do gene humano de distrofina por edição de gene e métodos de uso |
| EA201891419A1 (ru) * | 2015-12-15 | 2019-01-31 | Сарепта Терапьютикс, Инк. | Пептид-олигонуклеотидные конъюгаты |
| MD3464306T2 (ro) | 2016-05-24 | 2024-08-31 | Sarepta Therapeutics Inc | Procedee de preparare a oligomerilor morfolino fosforodiamidați |
| MA45154A (fr) | 2016-05-24 | 2019-04-10 | Sarepta Therapeutics Inc | Procédés de préparation d'oligomères |
| US11472824B2 (en) | 2016-05-24 | 2022-10-18 | Sarepta Therapeutics, Inc. | Processes for preparing phosphorodiamidate morpholino oligomers |
| MA45362A (fr) | 2016-05-24 | 2019-04-10 | Sarepta Therapeutics Inc | Procédés de préparation d'oligomères morpholino de phosphorodiamidate |
| CN109562123A (zh) * | 2016-05-24 | 2019-04-02 | 萨勒普塔医疗公司 | 磷酸二酰胺吗啉代寡聚物药物组合物 |
| US20190262375A1 (en) * | 2016-06-30 | 2019-08-29 | Sarepta Therapeutics, Inc. | Exon skipping oligomers for muscular dystrophy |
| CN106581043A (zh) * | 2016-10-24 | 2017-04-26 | 佳木斯大学 | 一种适用于杜氏肌营养不良症的药物制剂及其制备方法 |
| CA3044531A1 (en) * | 2016-11-28 | 2018-05-31 | The Board Of Regents Of The University Of Texas System | Prevention of muscular dystrophy by crispr/cpf1-mediated gene editing |
| ES2980686T3 (es) * | 2016-12-19 | 2024-10-02 | Sarepta Therapeutics Inc | Conjugados de oligómero de omisión de exones para distrofia muscular |
| EP3554553B1 (en) * | 2016-12-19 | 2022-07-20 | Sarepta Therapeutics, Inc. | Exon skipping oligomer conjugates for muscular dystrophy |
| KR20240006057A (ko) | 2016-12-19 | 2024-01-12 | 사렙타 쎄러퓨틱스 인코퍼레이티드 | 근육 이상증에 대한 엑손 스킵핑 올리고머 결합체 |
| CN110381980A (zh) * | 2017-01-06 | 2019-10-25 | 艾维迪提生物科学有限责任公司 | 核酸-多肽组合物以及诱导外显子跳读的方法 |
| US11622960B2 (en) | 2017-06-19 | 2023-04-11 | University Of Maryland, Baltimore | Microtubule polymerization inhibitor prodrugs and methods of using the same |
| CN111164211B (zh) | 2017-07-18 | 2024-08-02 | 杰特贝林基因治疗股份有限公司 | 用于治疗β-血红蛋白病的组合物和方法 |
| GB201711809D0 (en) | 2017-07-21 | 2017-09-06 | Governors Of The Univ Of Alberta | Antisense oligonucleotide |
| TW201919655A (zh) * | 2017-08-31 | 2019-06-01 | 美商薩羅塔治療公司 | 治療肌肉萎縮症的方法 |
| JP7441455B2 (ja) * | 2017-09-22 | 2024-03-01 | ザ リージェンツ オブ ザ ユニバーシティ オブ コロラド,ア ボディー コーポレイト | 筋ジストロフィーの処置のためのチオモルホリノオリゴヌクレオチド |
| JP2020537497A (ja) * | 2017-09-22 | 2020-12-24 | アビディティー バイオサイエンシーズ,インク. | エクソンスキッピングを誘発する核酸ポリペプチド組成物と方法 |
| US20200248178A1 (en) * | 2017-09-28 | 2020-08-06 | Sarepta Therapeutics, Inc. | Combination therapies for treating muscular dystrophy |
| BR112020007157A2 (pt) | 2017-10-13 | 2020-09-24 | Selecta Biosciences, Inc. | métodos e composições para a atenuação de respostas de igm antivetor de transferência viral |
| MX2020005860A (es) | 2017-12-06 | 2020-09-09 | Avidity Biosciences Inc | Composiciones y metodos de tratamiento de atrofia muscular y distrofia miotonica. |
| JP2021511803A (ja) * | 2018-01-31 | 2021-05-13 | ザ ボード オブ リージェンツ オブ ザ ユニバーシティー オブ テキサス システム | ヒト心筋細胞におけるジストロフィン変異を修正するための組成物および方法 |
| MA52148A (fr) * | 2018-03-16 | 2021-01-20 | Sarepta Therapeutics Inc | Peptides chimères pour administration d'antisens |
| EP3784248A4 (en) * | 2018-04-26 | 2022-08-10 | Sarepta Therapeutics, Inc. | EXON-SKIPPING OLIGOMERS AND OLIGOMER CONJUGATE FOR MUSCLE DYSTROPHY |
| US10765760B2 (en) * | 2018-05-29 | 2020-09-08 | Sarepta Therapeutics, Inc. | Exon skipping oligomer conjugates for muscular dystrophy |
| EP4219717A3 (en) * | 2018-06-13 | 2023-12-20 | Sarepta Therapeutics, Inc. | Exon skipping oligomers for muscular dystrophy |
| US20220251551A1 (en) * | 2018-06-13 | 2022-08-11 | Sarepta Therapeutics, Inc. | Exon skipping oligomers for muscular dystrophy |
| SG11202011554PA (en) | 2018-06-26 | 2020-12-30 | Nippon Shinyaku Co Ltd | Composition comprising antisense oligonucleotide and use thereof for treatment of duchenne muscular dystrophy |
| MX2021001281A (es) | 2018-08-02 | 2021-07-15 | Dyne Therapeutics Inc | Complejos dirigidos a músculos y sus usos para el tratamiento de distrofinopatías. |
| US11168141B2 (en) | 2018-08-02 | 2021-11-09 | Dyne Therapeutics, Inc. | Muscle targeting complexes and uses thereof for treating dystrophinopathies |
| CA3108289A1 (en) | 2018-08-02 | 2020-02-06 | Dyne Therapeutics, Inc. | Muscle targeting complexes and uses thereof for treating facioscapulohumeral muscular dystrophy |
| AU2019417697A1 (en) | 2018-12-23 | 2021-07-08 | Csl Behring L.L.C. | Haematopoietic stem cell-gene therapy for wiskott-aldrich syndrome |
| BR112021012318A2 (pt) | 2018-12-23 | 2022-01-18 | Csl Behring Llc | Células t doadoras com interruptor de eliminação |
| US20220098578A1 (en) | 2019-01-31 | 2022-03-31 | Bar Ilan University | Neoantigens created by aberrant-induced splicing and uses thereof in enhancing immunotherapy |
| SG11202108757XA (en) * | 2019-03-28 | 2021-10-28 | Sarepta Therapeutics Inc | Methods for treating muscular dystrophy with casimersen |
| BR112021023594A2 (pt) | 2019-05-28 | 2022-02-08 | Selecta Biosciences Inc | Métodos e composições para resposta imune de vetor de transferência antiviral atenuada |
| KR20220038771A (ko) * | 2019-08-02 | 2022-03-29 | 더 리서치 인스티튜트 앳 네이션와이드 칠드런스 하스피탈 | 엑손 44-표적화된 핵산 및 상기 핵산을 포함하는 디스트로핀-기반 근병증 치료용 재조합 아데노-관련 바이러스 |
| JP2023501222A (ja) * | 2019-10-30 | 2023-01-18 | 武田薬品工業株式会社 | オリゴヌクレオチドを検出するための方法 |
| MX2022007937A (es) * | 2019-12-26 | 2022-07-27 | Nippon Shinyaku Co Ltd | Acido nucleico antisentido que induce la omision del exon 50. |
| JP2023537798A (ja) | 2020-03-19 | 2023-09-06 | アビディティー バイオサイエンシーズ,インク. | 顔面肩甲上腕型筋ジストロフィーを処置するための組成物および方法 |
| EP4168425A4 (en) * | 2020-06-17 | 2024-06-19 | Stealth BioTherapeutics Inc. | METHODS AND COMPOSITIONS FOR THE TREATMENT OF MUSCULAR DYSTROPHY |
| AU2021294317A1 (en) | 2020-06-26 | 2023-02-23 | Csl Behring Llc | Donor T-cells with kill switch |
| US11771776B2 (en) | 2021-07-09 | 2023-10-03 | Dyne Therapeutics, Inc. | Muscle targeting complexes and uses thereof for treating dystrophinopathies |
| US11638761B2 (en) | 2021-07-09 | 2023-05-02 | Dyne Therapeutics, Inc. | Muscle targeting complexes and uses thereof for treating Facioscapulohumeral muscular dystrophy |
| WO2023044398A1 (en) * | 2021-09-16 | 2023-03-23 | Dyne Therapeutics, Inc. | Dosing of muscle targeting complexes for treating dystrophinopathies |
| KR20240055874A (ko) | 2021-09-16 | 2024-04-29 | 어비디티 바이오사이언시스 인크. | 안면견갑상완 근이영양증을 치료하는 조성물 및 방법 |
| US20230141563A1 (en) | 2021-10-12 | 2023-05-11 | Selecta Biosciences, Inc. | Methods and compositions for attenuating anti-viral transfer vector igm responses |
| CN118488959A (zh) | 2021-12-27 | 2024-08-13 | 日本新药株式会社 | 低聚核酸化合物的制造方法 |
| EP4215614A1 (en) | 2022-01-24 | 2023-07-26 | Dynacure | Combination therapy for dystrophin-related diseases |
| WO2023168427A1 (en) | 2022-03-03 | 2023-09-07 | Yale University | Compositions and methods for delivering therapeutic polynucleotides for exon skipping |
| US20230357437A1 (en) | 2022-03-09 | 2023-11-09 | Selecta Biosciences, Inc. | Immunosuppressants in combination with anti-igm agents and related dosing |
| IL315280A (en) * | 2022-03-17 | 2024-10-01 | Sarepta Therapeutics Inc | Morpholino phosphorodiamidate oligomer conjugates |
| US12071621B2 (en) | 2022-04-05 | 2024-08-27 | Avidity Biosciences, Inc. | Anti-transferrin receptor antibody-PMO conjugates for inducing DMD exon 44 skipping |
| WO2023250511A2 (en) | 2022-06-24 | 2023-12-28 | Tune Therapeutics, Inc. | Compositions, systems, and methods for reducing low-density lipoprotein through targeted gene repression |
| CN115820642B (zh) * | 2022-11-11 | 2023-10-10 | 昆明理工大学 | 一种用于治疗杜氏肌营养不良症的CRISPR-Cas9系统 |
Family Cites Families (143)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CH445129A (fr) | 1964-04-29 | 1967-10-15 | Nestle Sa | Procédé pour la préparation de composés d'inclusion à poids moléculaire élevé |
| US3459731A (en) | 1966-12-16 | 1969-08-05 | Corn Products Co | Cyclodextrin polyethers and their production |
| US3453257A (en) | 1967-02-13 | 1969-07-01 | Corn Products Co | Cyclodextrin with cationic properties |
| US3426011A (en) | 1967-02-13 | 1969-02-04 | Corn Products Co | Cyclodextrins with anionic properties |
| US3453259A (en) | 1967-03-22 | 1969-07-01 | Corn Products Co | Cyclodextrin polyol ethers and their oxidation products |
| US4235871A (en) | 1978-02-24 | 1980-11-25 | Papahadjopoulos Demetrios P | Method of encapsulating biologically active materials in lipid vesicles |
| US4458066A (en) | 1980-02-29 | 1984-07-03 | University Patents, Inc. | Process for preparing polynucleotides |
| US5190931A (en) | 1983-10-20 | 1993-03-02 | The Research Foundation Of State University Of New York | Regulation of gene expression by employing translational inhibition of MRNA utilizing interfering complementary MRNA |
| EP0639582B1 (en) | 1985-03-15 | 1998-09-16 | Antivirals Inc. | Polynucleotide assay reagent and method |
| US5034506A (en) | 1985-03-15 | 1991-07-23 | Anti-Gene Development Group | Uncharged morpholino-based polymers having achiral intersubunit linkages |
| US5521063A (en) | 1985-03-15 | 1996-05-28 | Antivirals Inc. | Polynucleotide reagent containing chiral subunits and methods of use |
| US5185444A (en) | 1985-03-15 | 1993-02-09 | Anti-Gene Deveopment Group | Uncharged morpolino-based polymers having phosphorous containing chiral intersubunit linkages |
| US5166315A (en) | 1989-12-20 | 1992-11-24 | Anti-Gene Development Group | Sequence-specific binding polymers for duplex nucleic acids |
| US5506337A (en) | 1985-03-15 | 1996-04-09 | Antivirals Inc. | Morpholino-subunit combinatorial library and method |
| US5217866A (en) | 1985-03-15 | 1993-06-08 | Anti-Gene Development Group | Polynucleotide assay reagent and method |
| US4737323A (en) | 1986-02-13 | 1988-04-12 | Liposome Technology, Inc. | Liposome extrusion method |
| KR0166088B1 (ko) | 1990-01-23 | 1999-01-15 | . | 수용해도가 증가된 시클로덱스트린 유도체 및 이의 용도 |
| US5149797A (en) | 1990-02-15 | 1992-09-22 | The Worcester Foundation For Experimental Biology | Method of site-specific alteration of rna and production of encoded polypeptides |
| US5138045A (en) | 1990-07-27 | 1992-08-11 | Isis Pharmaceuticals | Polyamine conjugated oligonucleotides |
| US20020123476A1 (en) | 1991-03-19 | 2002-09-05 | Emanuele R. Martin | Therapeutic delivery compositions and methods of use thereof |
| DE69329641T2 (de) | 1992-03-31 | 2001-06-21 | Abbott Laboratories, Abbott Park | Verfahren zur mehrfachen ligase-kettenreaktion |
| US5869252A (en) | 1992-03-31 | 1999-02-09 | Abbott Laboratories | Method of multiplex ligase chain reaction |
| US6806084B1 (en) | 1992-06-04 | 2004-10-19 | The Regents Of The University Of California | Methods for compositions for in vivo gene delivery |
| EP1251170A3 (en) | 1992-07-17 | 2002-10-30 | Ribozyme Pharmaceuticals, Inc. | Method and reagent for treatment of NF-kappaB dependent animal diseases |
| US5985558A (en) | 1997-04-14 | 1999-11-16 | Isis Pharmaceuticals Inc. | Antisense oligonucleotide compositions and methods for the inibition of c-Jun and c-Fos |
| CN1123038A (zh) | 1993-05-11 | 1996-05-22 | 北卡罗来纳大学查珀尔希尔分校 | 阻止异常剪接的反义寡核苷酸及其使用方法 |
| US5801154A (en) | 1993-10-18 | 1998-09-01 | Isis Pharmaceuticals, Inc. | Antisense oligonucleotide modulation of multidrug resistance-associated protein |
| US6391636B1 (en) | 1994-05-31 | 2002-05-21 | Isis Pharmaceuticals, Inc. | Antisense oligonucleotide modulation of raf gene expression |
| US5820873A (en) | 1994-09-30 | 1998-10-13 | The University Of British Columbia | Polyethylene glycol modified ceramide lipids and liposome uses thereof |
| US5753613A (en) | 1994-09-30 | 1998-05-19 | Inex Pharmaceuticals Corporation | Compositions for the introduction of polyanionic materials into cells |
| US5885613A (en) | 1994-09-30 | 1999-03-23 | The University Of British Columbia | Bilayer stabilizing components and their use in forming programmable fusogenic liposomes |
| IL115849A0 (en) | 1994-11-03 | 1996-01-31 | Merz & Co Gmbh & Co | Tangential filtration preparation of liposomal drugs and liposome product thereof |
| GB9510718D0 (en) | 1995-05-26 | 1995-07-19 | Sod Conseils Rech Applic | Antisense oligonucleotides |
| AU725262B2 (en) | 1996-02-14 | 2000-10-12 | Isis Pharmaceuticals, Inc. | Sugar-modified gapped oligonucleotides |
| AU2585197A (en) | 1996-03-20 | 1997-10-10 | Regents Of The University Of California, The | Antisense approach to gene inhibition |
| US6153436A (en) | 1997-01-10 | 2000-11-28 | The Board Of Trustees Of The University Of Arkansas | Method of gene delivery using wildtype adeno associated viral (AAV) vectors with insertions |
| WO1999042091A2 (en) | 1998-02-19 | 1999-08-26 | Massachusetts Institute Of Technology | Use of polycations as endosomolytic agents |
| US20030114401A1 (en) | 2001-12-06 | 2003-06-19 | Isis Pharmaceuticals Inc. | Antisense modulation of Ship-1 expression |
| GB9819999D0 (en) | 1998-09-14 | 1998-11-04 | Univ London | Treatment of cancer |
| US6210892B1 (en) | 1998-10-07 | 2001-04-03 | Isis Pharmaceuticals, Inc. | Alteration of cellular behavior by antisense modulation of mRNA processing |
| CA2350531A1 (en) | 1998-11-13 | 2000-05-25 | Smriti Iyengar | Method for treating pain |
| JP2002535015A (ja) | 1999-01-29 | 2002-10-22 | エイブイアイ バイオファーマ, インコーポレイテッド | 標的rnaを検出するための非侵襲性方法 |
| US20020049173A1 (en) | 1999-03-26 | 2002-04-25 | Bennett C. Frank | Alteration of cellular behavior by antisense modulation of mRNA processing |
| JP2000325085A (ja) | 1999-05-21 | 2000-11-28 | Masafumi Matsuo | デュシェンヌ型筋ジストロフィー治療剤 |
| WO2000078341A1 (en) | 1999-06-21 | 2000-12-28 | Murdoch Childrens Research Institute | A method for the prophylaxis and/or treatment of medical disorders |
| US7070807B2 (en) | 1999-12-29 | 2006-07-04 | Mixson A James | Branched histidine copolymers and methods for using same |
| US7163695B2 (en) | 1999-12-29 | 2007-01-16 | Mixson A James | Histidine copolymer and methods for using same |
| WO2001049775A2 (en) | 2000-01-04 | 2001-07-12 | Avi Biopharma, Inc. | Antisense antibacterial cell division composition and method |
| JP2004509604A (ja) | 2000-03-28 | 2004-04-02 | アイシス・ファーマシューティカルス・インコーポレーテッド | mRNAプロセッシングのアンチセンスモジュレーションによる、細胞行動の改変 |
| US6653467B1 (en) | 2000-04-26 | 2003-11-25 | Jcr Pharmaceutical Co., Ltd. | Medicament for treatment of Duchenne muscular dystrophy |
| DE60139394D1 (de) | 2000-04-28 | 2009-09-10 | Asklepios Biopharmaceutical In | Für das dystrophin-minigen kodierende dna-sequenzen und verfahren zu deren verwendung |
| US7115579B2 (en) | 2000-05-01 | 2006-10-03 | Idera Pharmaceuticals, Inc. | Modulation of oligonucleotide CpG-mediated immune stimulation by positional modification of nucleosides |
| US6784291B2 (en) | 2000-05-04 | 2004-08-31 | Avi Biopharma, Inc. | Splice-region antisense composition and method |
| US6727355B2 (en) | 2000-08-25 | 2004-04-27 | Jcr Pharmaceuticals Co., Ltd. | Pharmaceutical composition for treatment of Duchenne muscular dystrophy |
| JP4836366B2 (ja) | 2000-08-25 | 2011-12-14 | 雅文 松尾 | デュシェンヌ型筋ジストロフィー治療剤 |
| WO2002018656A2 (en) | 2000-08-30 | 2002-03-07 | Avi Biopharma, Inc. | Method for analysis of oligonucleotide analogs |
| US6559279B1 (en) | 2000-09-08 | 2003-05-06 | Isis Pharmaceuticals, Inc. | Process for preparing peptide derivatized oligomeric compounds |
| US20070037165A1 (en) | 2000-09-08 | 2007-02-15 | Applera Corporation | Polymorphisms in known genes associated with human disease, methods of detection and uses thereof |
| AU2001289085A1 (en) | 2000-09-20 | 2002-04-02 | Isis Pharmaceuticals, Inc. | Antisense modulation of flip-c expression |
| EP1191097A1 (en) | 2000-09-21 | 2002-03-27 | Leids Universitair Medisch Centrum | Induction of exon skipping in eukaryotic cells |
| US6689615B1 (en) | 2000-10-04 | 2004-02-10 | James Murto | Methods and devices for processing blood samples |
| JP3781687B2 (ja) | 2001-02-23 | 2006-05-31 | 松下電器産業株式会社 | 遺伝子診断装置及び遺伝子診断方法 |
| US6656732B1 (en) | 2001-05-18 | 2003-12-02 | Isis Pharmaceuticals, Inc. | Antisense inhibition of src-c expression |
| US20030224353A1 (en) | 2001-10-16 | 2003-12-04 | Stein David A. | Antisense antiviral agent and method for treating ssRNA viral infection |
| EP2305813A3 (en) | 2002-11-14 | 2012-03-28 | Dharmacon, Inc. | Fuctional and hyperfunctional sirna |
| US7655785B1 (en) | 2002-11-14 | 2010-02-02 | Rosetta Genomics Ltd. | Bioinformatically detectable group of novel regulatory oligonucleotides and uses thereof |
| US7314750B2 (en) | 2002-11-20 | 2008-01-01 | Affymetrix, Inc. | Addressable oligonucleotide array of the rat genome |
| US7250289B2 (en) | 2002-11-20 | 2007-07-31 | Affymetrix, Inc. | Methods of genetic analysis of mouse |
| ES2554660T3 (es) | 2002-11-25 | 2015-12-22 | Masafumi Matsuo | Fármacos de ácido nucleico ENA que modifican el corte y empalme en precursores de ARNm |
| WO2004083432A1 (en) | 2003-03-21 | 2004-09-30 | Academisch Ziekenhuis Leiden | Modulation of exon recognition in pre-mrna by interfering with the secondary rna structure |
| ES2500921T3 (es) | 2003-04-29 | 2014-10-01 | Sarepta Therapeutics, Inc. | Composiciones para potenciar el transporte y la eficacia antisentido de análogos de ácidos nucleicos en células |
| MXPA05012965A (es) * | 2003-06-02 | 2006-03-09 | Wyeth Corp | Uso de inhibidores de miostatina (gdf8) en conjuncion con corticoesteroides para el tratamiento de desordenes neuromusculares y composiciones farmaceuticas para ello. |
| ES2302898T3 (es) | 2003-07-11 | 2008-08-01 | Lbr Medbiotech B.V. | Transferencia de genes a celulas musculares mediada por el receptor de manosa-6-fosfato. |
| KR100943473B1 (ko) | 2003-08-05 | 2010-02-19 | 에이브이아이 바이오파마 인코포레이티드 | 올리고뉴클레오티드 유사체 및 플라비바이러스 감염을 치료하는 방법 |
| US20050048495A1 (en) | 2003-08-29 | 2005-03-03 | Baker Brenda F. | Isoform-specific targeting of splice variants |
| WO2005027833A2 (en) | 2003-09-12 | 2005-03-31 | Avi Biopharma, Inc. | Compound and method for treating androgen-independent prostate cancer |
| US20050171044A1 (en) | 2003-12-24 | 2005-08-04 | Stein David A. | Oligonucleotide compound and method for treating nidovirus infections |
| US20050288246A1 (en) | 2004-05-24 | 2005-12-29 | Iversen Patrick L | Peptide conjugated, inosine-substituted antisense oligomer compound and method |
| EP3808845A1 (en) | 2004-06-28 | 2021-04-21 | The University Of Western Australia | Antisense oligonucleotides for inducing exon skipping and methods of use thereof |
| FR2874384B1 (fr) | 2004-08-17 | 2010-07-30 | Genethon | Vecteur viral adeno-associe pour realiser du saut d'exons dans un gene codant une proteine a domaines dispensables |
| US8129352B2 (en) | 2004-09-16 | 2012-03-06 | Avi Biopharma, Inc. | Antisense antiviral compound and method for treating ssRNA viral infection |
| US20060148740A1 (en) | 2005-01-05 | 2006-07-06 | Prosensa B.V. | Mannose-6-phosphate receptor mediated gene transfer into muscle cells |
| US20120122801A1 (en) | 2005-01-05 | 2012-05-17 | Prosensa B.V. | Mannose-6-phosphate receptor mediated gene transfer into muscle cells |
| EP1855694B1 (en) | 2005-02-09 | 2020-12-02 | Sarepta Therapeutics, Inc. | Antisense composition for treating muscle atrophy |
| NO20051491L (no) | 2005-03-21 | 2006-09-22 | Norsk Hydro As | Fremgangsmate og anordning ved en gravitasjonsseparator. |
| EP1877555A2 (en) | 2005-04-22 | 2008-01-16 | Academisch Ziekenhuis Leiden | Modulation of exon recognition in pre-mrna by interfering with the binding of sr proteins and by interfering with secondary rna structure. |
| US8067571B2 (en) | 2005-07-13 | 2011-11-29 | Avi Biopharma, Inc. | Antibacterial antisense oligonucleotide and method |
| WO2007028065A2 (en) | 2005-08-30 | 2007-03-08 | Isis Pharmaceuticals, Inc. | Chimeric oligomeric compounds for modulation of splicing |
| WO2007030691A2 (en) | 2005-09-08 | 2007-03-15 | Avi Biopharma, Inc. | Antisense antiviral compound and method for treating picornavirus infection |
| US8501704B2 (en) | 2005-11-08 | 2013-08-06 | Sarepta Therapeutics, Inc. | Immunosuppression compound and treatment method |
| US20070105807A1 (en) | 2005-11-10 | 2007-05-10 | Sazani Peter L | Splice switch oligomers for TNF superfamily receptors and their use in treatment of disease |
| WO2007133812A2 (en) | 2005-12-30 | 2007-11-22 | Philadelphia Health & Education Corporation, D/B/A Drexel University College Of Medicine | Improved carriers for delivery of nucleic acid agents to cells and tissues |
| GB0602767D0 (en) * | 2006-02-10 | 2006-03-22 | Vastox Plc | Treatment of muscular dystrophy |
| AU2007297861A1 (en) | 2006-05-10 | 2008-03-27 | Avi Biopharma, Inc. | Oligonucleotide analogs having cationic intersubunit linkages |
| US8785407B2 (en) | 2006-05-10 | 2014-07-22 | Sarepta Therapeutics, Inc. | Antisense antiviral agent and method for treating ssRNA viral infection |
| US20070265215A1 (en) | 2006-05-11 | 2007-11-15 | Iversen Patrick L | Antisense restenosis composition and method |
| EP1857548A1 (en) | 2006-05-19 | 2007-11-21 | Academisch Ziekenhuis Leiden | Means and method for inducing exon-skipping |
| CA2691673A1 (en) | 2007-06-29 | 2009-01-08 | Avi Biopharma, Inc. | Tissue specific peptide conjugates and methods |
| US20100016215A1 (en) | 2007-06-29 | 2010-01-21 | Avi Biopharma, Inc. | Compound and method for treating myotonic dystrophy |
| CA2704049A1 (en) | 2007-10-26 | 2009-04-30 | Academisch Ziekenhuis Leiden | Means and methods for counteracting muscle disorders |
| US8076476B2 (en) | 2007-11-15 | 2011-12-13 | Avi Biopharma, Inc. | Synthesis of morpholino oligomers using doubly protected guanine morpholino subunits |
| US8299206B2 (en) | 2007-11-15 | 2012-10-30 | Avi Biopharma, Inc. | Method of synthesis of morpholino oligomers |
| WO2009101399A1 (en) | 2008-02-12 | 2009-08-20 | Isis Innovation Limited | Treatment of muscular dystrophy using peptide nucleic acid ( pna) |
| PT2108366E (pt) * | 2008-04-09 | 2011-11-10 | Santhera Pharmaceuticals Ch | Derivado de quinona 2,3-dimetoxi-5-metil-6-(10- hidroxidecil)-1,4-benzoquinona para o tratamento de doença respiratória em distrofia muscular |
| EP2119783A1 (en) | 2008-05-14 | 2009-11-18 | Prosensa Technologies B.V. | Method for efficient exon (44) skipping in Duchenne Muscular Dystrophy and associated means |
| US8084601B2 (en) | 2008-09-11 | 2011-12-27 | Royal Holloway And Bedford New College Royal Holloway, University Of London | Oligomers |
| US8871918B2 (en) | 2008-10-24 | 2014-10-28 | Sarepta Therapeutics, Inc. | Multiple exon skipping compositions for DMD |
| ES2562658T3 (es) | 2008-10-27 | 2016-03-07 | Biomarin Technologies B.V. | Procedimientos y medios para el salto eficiente del exón 45 en el pre-ARNm de la distrofia muscular de Duchenne |
| WO2010080554A1 (en) | 2008-12-17 | 2010-07-15 | Avi Biopharma, Inc. | Antisense compositions and methods for modulating contact hypersensitivity or contact dermatitis |
| WO2010115993A1 (en) | 2009-04-10 | 2010-10-14 | Association Institut De Myologie | Tricyclo-dna antisense oligonucleotides, compositions, and methods for the treatment of disease |
| AU2010239779A1 (en) | 2009-04-24 | 2011-11-17 | Prosensa Technologies B.V. | Oligonucleotide comprising an inosine for treating DMD |
| US9034838B2 (en) | 2009-05-25 | 2015-05-19 | Universita Degli Studi Di Roma “La Sapienza” | miR-31 in duchenne muscular dystrophy therapy |
| EP2258863A1 (en) | 2009-05-25 | 2010-12-08 | Universita'Degli Studi di Roma "La Sapienza" | miRNA biomarkers for the diagnosis of duchenne muscular dystrophy progression, for monitoring therapeutic interventions, and as therapeutics |
| US20110269665A1 (en) | 2009-06-26 | 2011-11-03 | Avi Biopharma, Inc. | Compound and method for treating myotonic dystrophy |
| ITTO20090487A1 (it) | 2009-06-26 | 2010-12-27 | Univ Ferrara | Oligonucleotidi antisenso atti ad indurre lo skipping esonico e loro impiego come medicamento per il trattamento della distrofia muscolare di duchenne (dmd) |
| EP2473607A2 (en) | 2009-08-31 | 2012-07-11 | INSERM - Institut National de la Santé et de la Recherche Médicale | Exon skipping therapy for functional amelioration of semi functional dystrophin in becker and duchenne muscular dystrophy |
| IT1397011B1 (it) | 2009-10-14 | 2012-12-20 | Univ Ferrara | Nanoparticella del tipo core-shell idonea per la veicolazione di oligonucleotidi terapeutici in tessuti bersaglio e suo impiego per la preparazione di un medicamento per il trattamento della distrofia muscolare di duchenne. |
| KR102581868B1 (ko) | 2009-11-12 | 2023-10-04 | 더 유니버시티 오브 웨스턴 오스트레일리아 | 안티센스 분자 및 이를 이용한 질환 치료방법 |
| TWI495473B (zh) | 2009-11-13 | 2015-08-11 | Sarepta Therapeutics Inc | 反義抗病毒化合物及治療流感病毒感染的方法 |
| US9050373B2 (en) | 2010-05-13 | 2015-06-09 | The Charlotte-Mecklenburg Hospital Authority | Pharmaceutical compositions comprising antisense oligonucleotides and methods of using same |
| JP2013530154A (ja) | 2010-05-28 | 2013-07-25 | サレプタ セラピューティクス, インコーポレイテッド | 修飾されたサブユニット間結合および/または末端基を有するオリゴヌクレオチドアナログ |
| US9241929B2 (en) | 2010-06-28 | 2016-01-26 | Masatoshi Hagiwara | Prophylactic or ameliorating agent for genetic diseases |
| TWI541024B (zh) | 2010-09-01 | 2016-07-11 | 日本新藥股份有限公司 | 反義核酸 |
| RS55610B1 (sr) | 2010-09-30 | 2017-06-30 | Nippon Shinyaku Co Ltd | Derivat morfolino nukleinske kiseline |
| CN103501793A (zh) | 2011-02-08 | 2014-01-08 | 夏洛特-梅克伦堡医院(商业用名:卡罗来纳保健系统) | 反义寡核苷酸 |
| US9161948B2 (en) | 2011-05-05 | 2015-10-20 | Sarepta Therapeutics, Inc. | Peptide oligonucleotide conjugates |
| IL273838B (en) | 2011-05-05 | 2022-09-01 | Sarepta Therapeutics Inc | Peptide and oligonucleotide conjugates, preparations containing them and their uses |
| WO2013033407A2 (en) | 2011-08-30 | 2013-03-07 | The Regents Of The University Of California | Identification of small molecules that enhance therapeutic exon skipping |
| US20140080896A1 (en) | 2011-08-30 | 2014-03-20 | The Regents Of The University Of California | Identification of small molecules that facilitate therapeutic exon skipping |
| PT2754440T (pt) * | 2011-09-05 | 2019-07-11 | Mitsubishi Tanabe Pharma Corp | Agente medicinal para tratar a esclerose lateral amiotrófica ou prevenir a progressão de uma fase da esclerose lateral amiotrófica |
| EP2581448B1 (en) | 2011-10-13 | 2015-01-28 | Association Institut de Myologie | Tricyclo-phosphorothioate DNA |
| CN110055243B (zh) | 2011-12-28 | 2024-03-26 | 日本新药株式会社 | 反义核酸 |
| CN104203289B (zh) | 2012-01-27 | 2020-11-03 | 比奥马林技术公司 | 用于治疗杜兴型肌营养不良症和贝克型肌营养不良症的具有改善特性的rna调节性寡核苷酸 |
| JP6542662B2 (ja) | 2012-03-20 | 2019-07-10 | サレプタ セラピューティクス, インコーポレイテッド | オリゴヌクレオチドアナログのボロン酸結合体 |
| JP6460983B2 (ja) | 2012-07-03 | 2019-01-30 | バイオマリン テクノロジーズ ベー.フェー. | 筋ジストロフィー患者の治療のためのオリゴヌクレオチド |
| CN111440796A (zh) * | 2012-12-20 | 2020-07-24 | 萨雷普塔医疗公司 | 用于治疗肌营养不良症的改良的外显子跳跃组合物 |
| CN110066793A (zh) | 2013-03-14 | 2019-07-30 | 萨勒普塔医疗公司 | 用于治疗肌肉萎缩的外显子跳跃组合物 |
| CA2906209A1 (en) | 2013-03-14 | 2014-09-25 | Sarepta Therapeutics, Inc. | Exon skipping compositions for treating muscular dystrophy targeting the annealing site h44a (-07+15) |
| JP2016516066A (ja) | 2013-03-15 | 2016-06-02 | サレプタ セラピューティクス, インコーポレイテッド | 筋ジストロフィを処置するための改善された組成物 |
| JP6461917B2 (ja) | 2013-04-20 | 2019-01-30 | リサーチ インスティチュート アット ネイションワイド チルドレンズ ホスピタル | エクソン2標的U7snRNAポリヌクレオチド構築物の組換えアデノ随伴ウイルスによる送達 |
| AU2014274840B2 (en) * | 2013-06-05 | 2020-03-12 | Duke University | RNA-guided gene editing and gene regulation |
| CN108738310A (zh) | 2015-09-30 | 2018-11-02 | 萨勒普塔医疗公司 | 用于治疗肌营养不良的方法 |
| JP7441455B2 (ja) * | 2017-09-22 | 2024-03-01 | ザ リージェンツ オブ ザ ユニバーシティ オブ コロラド,ア ボディー コーポレイト | 筋ジストロフィーの処置のためのチオモルホリノオリゴヌクレオチド |
-
2014
- 2014-03-14 JP JP2016503163A patent/JP2016516066A/ja not_active Withdrawn
- 2014-03-14 CN CN201480026195.5A patent/CN105307723A/zh active Pending
- 2014-03-14 AU AU2014233456A patent/AU2014233456B2/en active Active
- 2014-03-14 EP EP14722043.8A patent/EP2968991A2/en not_active Withdrawn
- 2014-03-14 WO PCT/US2014/029610 patent/WO2014144978A2/en active Application Filing
- 2014-03-14 KR KR1020207034743A patent/KR20200139271A/ko not_active Application Discontinuation
- 2014-03-14 US US14/213,629 patent/US20140329762A1/en not_active Abandoned
- 2014-03-14 EP EP19207586.9A patent/EP3662912A1/en active Pending
- 2014-03-14 US US14/214,567 patent/US9506058B2/en active Active
- 2014-03-14 CN CN202110759294.6A patent/CN113633787A/zh active Pending
- 2014-03-14 MX MX2015013117A patent/MX2015013117A/es active IP Right Grant
- 2014-03-14 BR BR112015022998A patent/BR112015022998A2/pt not_active Application Discontinuation
- 2014-03-14 NZ NZ631289A patent/NZ631289A/en unknown
- 2014-03-14 KR KR1020157029546A patent/KR20150133768A/ko not_active Application Discontinuation
- 2014-03-14 CA CA2906812A patent/CA2906812A1/en active Pending
- 2014-03-14 NZ NZ732507A patent/NZ732507A/en unknown
- 2014-03-14 EA EA201591792A patent/EA201591792A1/ru unknown
-
2015
- 2015-09-10 IL IL24155815A patent/IL241558B/en active IP Right Grant
- 2015-09-15 MX MX2020004986A patent/MX2020004986A/es unknown
-
2016
- 2016-07-12 HK HK16108175.2A patent/HK1220154A1/zh unknown
- 2016-11-22 US US15/359,152 patent/US10337003B2/en active Active
-
2017
- 2017-05-24 US US15/604,335 patent/US10364431B2/en active Active
-
2019
- 2019-02-22 JP JP2019030772A patent/JP2019108349A/ja active Pending
- 2019-05-20 AU AU2019203505A patent/AU2019203505A1/en not_active Abandoned
- 2019-05-24 US US16/422,241 patent/US20190359982A1/en not_active Abandoned
- 2019-06-07 US US16/434,799 patent/US20200040337A1/en not_active Abandoned
- 2019-10-06 IL IL26984519A patent/IL269845A/en unknown
-
2020
- 2020-10-29 AU AU2020260492A patent/AU2020260492A1/en not_active Abandoned
- 2020-11-23 IL IL278912A patent/IL278912A/en unknown
-
2021
- 2021-10-21 US US17/506,846 patent/US20220033820A1/en not_active Abandoned
-
2022
- 2022-01-03 US US17/567,252 patent/US20220403385A1/en not_active Abandoned
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| KR20150133768A (ko) | 근육 이영양증의 치료를 위한 개선된 조성물 | |
| EP3133160B1 (en) | Exon skipping compositions for dmd | |
| EP3858993A1 (en) | Compositions and methods for treating duchenne muscular dystrophy and related disorders | |
| JP2019050834A (ja) | 筋ジストロフィを処置するための改善されたエキソンスキッピング組成物 | |
| KR20150133751A (ko) | 근육 이영양증의 치료를 위한 엑손 스키핑 조성물 | |
| US20220193246A1 (en) | Compositions for treating muscular dystrophy | |
| US20210145852A1 (en) | Combination Therapies for Treating Muscular Dystrophy | |
| AU2019200666A1 (en) | Multiple exon skipping compositions for DMD |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| E902 | Notification of reason for refusal | ||
| A107 | Divisional application of patent | ||
| E601 | Decision to refuse application |