BRPI1006448B1 - Anticorpo antagonista anti-fgfr3, anticorpo monoclonal, polinucleotídeo, vetor, microorganismo transgênico, método para produção de um anticorpo anti-fgfr3, formulação farmacêutica e usos do anticorpo antagonista anti-fgfr3 - Google Patents
Anticorpo antagonista anti-fgfr3, anticorpo monoclonal, polinucleotídeo, vetor, microorganismo transgênico, método para produção de um anticorpo anti-fgfr3, formulação farmacêutica e usos do anticorpo antagonista anti-fgfr3 Download PDFInfo
- Publication number
- BRPI1006448B1 BRPI1006448B1 BRPI1006448-6A BRPI1006448A BRPI1006448B1 BR PI1006448 B1 BRPI1006448 B1 BR PI1006448B1 BR PI1006448 A BRPI1006448 A BR PI1006448A BR PI1006448 B1 BRPI1006448 B1 BR PI1006448B1
- Authority
- BR
- Brazil
- Prior art keywords
- antibody
- fgfr3
- seq
- hvr
- cancer
- Prior art date
Links
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/2863—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against receptors for growth factors, growth regulators
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/395—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/395—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum
- A61K39/39533—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum against materials from animals
- A61K39/3955—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum against materials from animals against proteinaceous materials, e.g. enzymes, hormones, lymphokines
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/395—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum
- A61K39/39533—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum against materials from animals
- A61K39/39558—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum against materials from animals against tumor tissues, cells, antigens
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P19/00—Drugs for skeletal disorders
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P37/00—Drugs for immunological or allergic disorders
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P43/00—Drugs for specific purposes, not provided for in groups A61P1/00-A61P41/00
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/30—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants from tumour cells
- C07K16/3038—Kidney, bladder
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/30—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants from tumour cells
- C07K16/3061—Blood cells
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/505—Medicinal preparations containing antigens or antibodies comprising antibodies
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/20—Immunoglobulins specific features characterized by taxonomic origin
- C07K2317/24—Immunoglobulins specific features characterized by taxonomic origin containing regions, domains or residues from different species, e.g. chimeric, humanized or veneered
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/30—Immunoglobulins specific features characterized by aspects of specificity or valency
- C07K2317/34—Identification of a linear epitope shorter than 20 amino acid residues or of a conformational epitope defined by amino acid residues
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/52—Constant or Fc region; Isotype
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/55—Fab or Fab'
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/56—Immunoglobulins specific features characterized by immunoglobulin fragments variable (Fv) region, i.e. VH and/or VL
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/56—Immunoglobulins specific features characterized by immunoglobulin fragments variable (Fv) region, i.e. VH and/or VL
- C07K2317/565—Complementarity determining region [CDR]
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/56—Immunoglobulins specific features characterized by immunoglobulin fragments variable (Fv) region, i.e. VH and/or VL
- C07K2317/567—Framework region [FR]
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/73—Inducing cell death, e.g. apoptosis, necrosis or inhibition of cell proliferation
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/73—Inducing cell death, e.g. apoptosis, necrosis or inhibition of cell proliferation
- C07K2317/732—Antibody-dependent cellular cytotoxicity [ADCC]
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/75—Agonist effect on antigen
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/76—Antagonist effect on antigen, e.g. neutralization or inhibition of binding
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/77—Internalization into the cell
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/90—Immunoglobulins specific features characterized by (pharmaco)kinetic aspects or by stability of the immunoglobulin
- C07K2317/92—Affinity (KD), association rate (Ka), dissociation rate (Kd) or EC50 value
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/30—Non-immunoglobulin-derived peptide or protein having an immunoglobulin constant or Fc region, or a fragment thereof, attached thereto
Abstract
"anticorpo antagonista anti-fgfr3, polinucleotídeo, vetor, micro-organismo transgênico, método para produção de um anticorpo anti-fgfr3, formulação farmacêutica e usos do anticorpo antagonista anti-fgfr3" a presente invenção está relacionada a anticorpos para fgfr3, composições compreendendo os mesmos e métodos de uso destes anticorpos.
Description
[001] O presente pedido reivindica a prioridade ao pedido de Patente Norte Americano número 61/163.222, depositado em 25 de março de 2009, cujo conteúdo é incorporado ao presente pela referência.
[002] A presente invenção está relacionada de maneira geral ao campo da biologia molecular. Mais especificamente, a presente invenção refere- se a anticorpos anti-FGFR3 e ao uso dos mesmos.
[003] Fatores de crescimento fibroblástico (FGF) e seus receptores (FGFRs) desempenham um papel crítico durante o desenvolvimento embrionário, na homeostase dos tecidos e no metabolismo (1-3). Nos humanos, existem 22 FGFs (FGF1-14, FGF16-23) e quatro receptores de FGF com domínio tirosina quinase (FGFR1-4). Os FGFRs consistem de uma região de ligação ao ligante extracelular, com dois ou três domínios semelhantes à imunoglobulina (immunoglobulin-like) (IgD1-3), uma região transmembrana de passagem única, e uma região citoplasmática, domínio tirosina quinase separado. Cada FGFR1, 2 e 3 possuem duas principais isoformas com splicing alternativo, designadas IIIb e IIIc. Estas isoformas diferem em cerca de 50 aminoácidos na segunda metade do IgD3, e possuem distribuição tecidual e especificidade do ligante distinta. De modo geral a isoforma IIIb é encontrada em células epiteliais, enquanto a forma IIIc é expressa em células mesenquimais. Após a ligação do FGF em conjunto com proteoglicanos de heparan sulfato, os FGFRs dimerizam e tornar-se fosforilados nos resíduos de tirosina específicos. Isso facilita o recrutamento de importantes proteínas adaptadoras, como o substrato de FGFR 2 α (FRS2α), levando à ativação de diversas cascatas de sinalização, incluindo as vias da proteína quinase ativada por mitógeno (MAPK) e PI3K-AKT (1, 3, 4). Consequentemente, os FGFs e seus receptores cognatos regulam uma ampla variedade de processos celulares, incluindo a proliferação, diferenciação, migração e sobrevivência, de forma dependente do contexto.
[004] Os FGFRs ativados de maneira aberrante têm sido implicados com doenças malignas específicas de humanos (1, 5). Em particular, a translocação cromossômica t (4;14) (p16.3;q32) ocorre em cerca de 15-20% dos pacientes com mieloma múltiplo, levando a superexpressão de FGFR3 e correlacionando-se com menor sobrevida global (6-9). O FGFR3 está envolvido também na conferência da quimiorresistência para linhagens de células de mieloma em cultura (10), o que é consistente com a má resposta clínica dos pacientes com t (4; 14) à quimioterapia convencional (8). A superexpressão do FGFR3 ativado de maneira mutacional é suficiente para induzir a transformação oncogênica de células hematopoiéticas e fibroblastos (11-14, 15), em modelos de camundongos transgênicos (16), e modelos murinos de transplante de medula óssea (16, 17). Assim, foi proposto o FGFR3 como um alvo terapêutico em potencial para o tratamento do mieloma múltiplo. De fato, vários inibidores do tipo pequenas moléculas direcionados para FGFRs, embora não seletivos para FGFR3 e possuindo atividade inibitória cruzada para algumas outras quinases, demonstraram citotoxicidade contra células de mieloma FGFR3 positivas em cultura de células e modelos de camundongos (18-22).
[005] A superexpressão de FGFR3 também foi documentada em uma alta fração de cânceres de bexiga (23, 24). Além disso, foram identificadas mutações ativadoras somáticas no FGFR3 em 60-70% dos carcinomas papilares e 16-20% dos carcinomas músculo-invasivos de bexiga (24, 25). Em experimentos de cultura celular, o RNA de interferência (11, 26) ou um fragmento de anticorpo FGFR3 Fv de cadeia única inibiu a proliferação de células de câncer de bexiga (27). Um estudo recente demonstrou que um conjugado anticorpo FGFR3-toxina atenua o crescimento do xenoenxerto de uma linhagem de células de câncer de bexiga por meio da entrega da toxina nos tumores mediada pelo FGFR3 (28). No entanto, não fica claro se a sinalização FGFR3 é de fato um fator oncogênico no crescimento in vivo de tumores de bexiga. Além disso, o potencial terapêutico no tratamento direcionado para FGFR3 no câncer de bexiga não foi definido com base em modelos in vivo. Publicações relacionadas com FGFR3 e anticorpos anti-FGFR3 incluem; Patente US 2005/0147612; Rauchenberger et al., J Biol Chem 278 (40):38194-38205 (2003); publicação WO2006/048877; Martinez-Torrecuadrada et al., (2008) Mol Cancer Ther 7 (4) 862-873; WO2007/144893; Trudel et al. (2006) 107(10): 4039-4046; Martinez- Torrecuadrada et al. (2005) Clin Cancer Res 11 (17): 6280-6290; Gomez-Roman et al. (2005) Clin Cancer Res 11:459-465; Direnzo, R (2007) Proceedings of AACR Annual Meeting, Resumo N°. 2080; publicação WO2010/002862.
[006] Está claro que ainda há a necessidade de agentes que possuam atributos clínicos que são ideais para o desenvolvimento como agentes terapêuticos. A presente invenção descrita no presente atende a essa necessidade e oferece outros benefícios.
[007] Todas as referências citadas no presente, incluindo patentes e publicações, são incorporadas ao presente pela referência em sua totalidade.
[008] A presente invenção é baseada em parte, na identificação de uma variedade de agentes ligantes de FGFR3 (tais como anticorpos e fragmentos destes). O FGFR3 apresenta-se como um importante e vantajoso alvo terapêutico, e a presente invenção fornece composições e métodos baseados na ligação de agentes ao FGFR3. Agentes ligantes de FGFR3 da presente invenção, conforme descrito no presente, fornecem importantes agentes terapêuticos e de diagnóstico para uso nas condições patológicas alvos associadas com a expressão e/ou atividade das vias de sinalização de FGFR3. Consequentemente, a presente invenção fornece métodos, composições, kits e artigos manufaturados relacionados com ligantes de FGFR3.
[009] A presente invenção fornece anticorpos que se ligam a FGFR3. Em um aspecto, a presente invenção apresente um anticorpo isolado que se liga um FGFR3. Em alguns exemplos de realização, o anticorpo se liga a um FGFR3 isoforma IIIb e/ou um FGFR3 isoforma IIIc. Em alguns exemplos de realização, o anticorpo se liga a um FGFR3 mutado (por exemplo, um ou mais dos FGFR3 IIIb R248C, S249C, G372C, Y375C, K652E, e/ou um ou mais dos FGFR3 IIIc R248C, S249C, G370C, Y373C, K650E). Em alguns exemplos de realização, o anticorpo se liga a um FGFR3 monomérico (por exemplo FGFR3 monomérico isoformas IIIb e/ou IIIc). Em alguns exemplos de realização, o anticorpo promove formação de FGFR3 monomérico, tal como pela estabilização da forma monomérica de FGFR3 em relação à forma dimérica de FGFR3.
[010] Em um aspecto, a presente invenção fornece um anticorpo anti-FGFR3 isolado, onde uma forma IgG do anticorpo de comprimento total se liga ao FGFR3 humano com uma Kd de 1 x 10-7, ou mais forte. Conforme está bem estabelecido no estado da técnica, a afinidade de ligação de um ligante ao seu receptor pode ser determinada utilizando qualquer um dentre uma série de ensaios, e pode ser expresso em termos de uma variedade de valores quantitativos. Assim, em um exemplo de realização, a afinidade é expressa como valores Kd e reflete a afinidade de ligação intrínseca (por exemplo, com efeitos de avidez minimizados). Geralmente, e preferencialmente, a afinidade de ligação é mensurada de maneira in vitro, seja em um modo sem células ou em modo associado à célula. Qualquer um dentre uma série de ensaios conhecidos no estado da técnica, incluindo aquelas descritas no presente, podem ser usadas para se obter as medidas de afinidade, incluindo, por exemplo, Biacore, radioimunoensaio (RIA) e ELISA. Em alguns exemplos de realização, a forma IgG de comprimento total do anticorpo se liga ao FGFR3 humano com uma Kd de 1 x 10-8, ou mais forte, com uma Kd de 1 x 10-9 ou mais forte, ou com uma Kd de 1 x 10-10 ou mais forte.
[011] De modo geral, os anticorpos anti-FGFR3 da presente invenção são anticorpos antagonistas. Desse modo, em um aspecto, os anticorpos anti-FGFR3 inibem a atividade de FGFR3 (por exemplo, a atividade de FGFR3-IIIb e/ou FGFR3-IIIc). Em alguns exemplos de realização, o anticorpo anti-FGFR3 (geralmente na forma bivalente) não possui função agonista de FGFR3 substancial. Em alguns exemplos de realização, o anticorpo antagonista anti-FGFR3 (geralmente na forma bivalente) possui pouca ou nenhuma função agonista de FGFR3. Em um exemplo de realização, um anticorpo da presente invenção (geralmente na forma bivalente) não exibe um nível de atividade agonista para FGFR3 que está acima do nível do fundo (background) que é de significância estatística.
[012] Em um aspecto, a ligação do anticorpo a um FGFR3 pode inibir a dimerização do receptor com outra unidade do receptor, de modo que a ativação do receptor é inibida (devido, pelo menos em parte, à falta de dimerização do receptor). A inibição pode ser direta ou indireta.
[013] Em um aspecto, a presente invenção fornece anticorpos anti-FGFR3 que não possuem atividade apoptótica substancial (por exemplo, não induzem a apoptose de uma célula, por exemplo, uma célula de carcinoma de células de transição ou uma célula de mieloma múltiplo, tal como uma célula de mieloma múltiplo compreendendo uma translocação de FGFR3, como na translocação t(4, 14)). Em alguns exemplos de realização, o anticorpo anti- FGFR3 possui pouca ou nenhuma função apoptótica. Em alguns exemplos de realização, os anticorpos FGFR3 não exibem uma função apoptótica que está acima do nível do fundo (background) que é de significância estatística.
[014] Em um aspecto, a presente invenção fornece anticorpos anti-FGFR3 que não induzem a uma regulação negativa (down-regulation) substancial de FGFR3. Em alguns exemplos de realização, o anticorpo anti- FGFR3 induz pouca ou nenhuma regulação negativa (down-regulation) do receptor. Em alguns exemplos de realização, os anticorpos FGFR3 não exibem uma regulação negativa (down-regulation) que está acima do nível de fundo (background) que é de significância estatística.
[015] Em um aspecto, a presente invenção fornece anticorpos anti-FGFR3 que possuem função efetora. Em um exemplo de realização, a função efetora compreende a citotoxicidade mediada por células dependente de anticorpos (ADCC). Em um exemplo de realização, o anticorpo anti-FGFR3 (em algumas realizações, um anticorpo anti-FGFR3 nu) é capaz de matar uma célula, e em alguns exemplos de realização, células de mieloma múltiplo (por exemplo, células de mieloma múltiplo compreendendo uma translocação, por exemplo, um translocação t(4, 14)). Em alguns exemplos de realização, o anticorpo anti- FGFR3 é capaz de matar uma célula que expressa cerca de 10.000 moléculas de FGFR3 por célula ou mais (como cerca de 11.000, cerca de 12.000, cerca de 13.000, cerca de 14.000, cerca de 15.000, cerca de 16.000, cerca de 17.000, cerca de 18.000 ou mais moléculas de FGFR3 por célula). Em outros exemplos de realização, a célula expressa cerca de 2000, cerca de 3000, cerca de 4000, cerca de 5000, cerca de 6000, cerca de 7000, cerca de 8000, ou mais moléculas de FGFR3 por célula.
[016] Em um aspecto, o anticorpo anti-FGFR3 da presente invenção inibe a atividade constitutiva de FGFR3. Em alguns exemplos de realização, a atividade do FGFR3 constitutiva é atividade constitutiva do FGFR3 dependente de ligante. Em alguns exemplos de realização, a atividade do FGFR3 constitutiva é atividade constitutiva do FGFR3 independente de ligante.
[017] Em um aspecto, o anticorpo anti-FGFR3 inibe o FGFR3 que compreende uma mutação correspondente ao FGFR3-IIIbR248C. Conforme usado no presente o termo “compreende uma mutação correspondente ao FGFR3-IIIbR248C” é entendido por abranger FGFR3-IIIbR248C e FGFR3- IIIcR248C, bem como formas de FGFR3 adicionais compreendendo uma mutação de R para C em uma posição correspondente ao do FGFR3-IIIb R248. Um técnico hábil no assunto entende como alinhar sequências FGFR3 a fim identificar os resíduos correspondentes entre as sequências de FGFR3 respectivas, por exemplo, alinhar uma sequência FGFR3-IIIc com uma sequência FGFR3-IIIb para identificar à posição no FGFR3 correspondente a posição R248 no FGFR3-IIIb. Em alguns exemplos de realização, o anticorpo anti-FGFR3 inibe FGFR3-IIIbR248C e/ou FGFR3-IIIcR248C.
[018] Em um aspecto, o anticorpo anti-FGFR3 inibe o FGFR3 que compreende uma mutação correspondente ao FGFR3-IIIbK652E. Por conveniência, o termo “compreende uma mutação correspondente ao FGFR3- IIIbK652E” é entendido por abranger FGFR3-IIIbK652E e FGFR3-IIIcK650E, bem como formas de FGFR3 adicionais compreendendo uma mutação de K para E em uma posição correspondente ao do FGFR3-IIIb K652. Um técnico hábil no assunto entende como alinhar sequências FGFR3 a fim identificar os resíduos correspondentes entre as sequências de FGFR3 respectivas, por exemplo, alinhar uma sequência FGFR3-IIIc com uma sequência FGFR3-IIIb para identificar à posição no FGFR3 correspondente a posição K652 no FGFR3-IIIb. Em alguns exemplos de realização, o anticorpo anti-FGFR3 inibe FGFR3- IIIbK652E e/ou FGFR3-IIIcK650E.
[019] Em um aspecto, o anticorpo anti-FGFR3 inibe o FGFR3 que compreende uma mutação correspondente ao FGFR3-IIIbS249C. Por conveniência, o termo “compreende uma mutação correspondente ao FGFR3- IIIbS249C” é entendido por abranger FGFR3-IIIbS249C e FGFR3-IIIcS249C, bem como formas de FGFR3 adicionais compreendendo uma mutação de S para C em uma posição correspondente ao do FGFR3-IIIb S249. Em alguns exemplos de realização, o anticorpo anti-FGFR3 inibe FGFR3-IIIbS249C e/ou FGFR3-IIIcS249C.
[020] Em um aspecto, o anticorpo anti-FGFR3 inibe o FGFR3 que compreende uma mutação correspondente ao FGFR3-IIIbG372C. Por conveniência, o termo “compreende uma mutação correspondente ao FGFR3- IIIbG372C” é entendido por abranger FGFR3-IIIbG372C e FGFR3-IIIcG370C, bem como formas de FGFR3 adicionais compreendendo uma mutação de G para C em uma posição correspondente ao do FGFR3-IIIb G372. Em alguns exemplos de realização, o anticorpo anti-FGFR3 inibe FGFR3-IIIbG372C e/ou FGFR3- IIIcG370C.
[021] Em um aspecto, o anticorpo anti-FGFR3 inibe o FGFR3 que compreende uma mutação correspondente ao FGFR3-IIIbY375C. Por conveniência, o termo “compreende uma mutação correspondente ao FGFR3- IIIbY375C” é entendido por abranger FGFR3-IIIbY375C e FGFR3-IIIcY373C, bem como formas de FGFR3 adicionais compreendendo uma mutação de S para C em uma posição correspondente ao do FGFR3-IIIb S249 (Y375). Em alguns exemplos de realização, o anticorpo anti-FGFR3 inibe FGFR3-IIIbY375C e/ou FGFR3-IIIcY373C.
[022] Em um aspecto, os anticorpos anti-FGFR3 inibem (a) e FGFR3-IIIbK652E e (b) um ou mais dos FGFR3-IIIbR248C, FGFR3-IIIbY375C, FGFR3- IIIbS249C e FGFR3-IIIbG372C.
[023] Em um aspecto, os anticorpos anti-FGFR3 inibem (a) e FGFR3-IIIcK650E e (b) um ou mais dos FGFR3-IIIcR248C, FGFR3-IIIcY373C, FGFR3- IIIcS249C e FGFR3-IIIcG370C.
[024] Em um aspecto, os anticorpos anti-FGFR3 inibem (a) e FGFR3-IIIbR248C e (b) um ou mais dos FGFR3-IIIbK652E, FGFR3-IIIbY375C, FGFR3- IIIbS249C e FGFR3-IIIbG372C.
[025] Em um aspecto, os anticorpos anti-FGFR3 inibem (a) e FGFR3-IIIcR248C e (b) um ou mais dos FGFR3-IIIcK650E, FGFR3-IIIcY373C, FGFR3- IIIcS249C e FGFR3-IIIcG370C.
[026] Em um aspecto, os anticorpos anti-FGFR3 inibem (a) e FGFR3-IIIbG372C e (b) um ou mais dos FGFR3-IIIbK652E, FGFR3-IIIbY375C, FGFR3- IIIbS249C e FGFR3-IIIbR248C.
[027] Em um aspecto, os anticorpos anti-FGFR3 inibem (a) e FGFR3-IIIcG370C e (b) um ou mais dos FGFR3-IIIcK650E, FGFR3-IIIcY373C, FGFR3- IIIcS249C e FGFR3-IIIcR248C.
[028] Em um aspecto, os anticorpos anti-FGFR3 inibem FGFR3- IIIbR248C, FGFR3-IIIbK652E, FGFR3-IIIbY375C, FGFR3-IIIbS249C e FGFR3-IIIbG372C.
[029] Em um aspecto, os anticorpos anti-FGFR3 inibem FGFR3- IIIcR248C, FGFR3-IIIcK650E, FGFR3-IIIcY373C, FGFR3-IIIcS249C e FGFR3-IIIcG370C.
[030] Em um aspecto, a presente invenção fornece um anticorpo anti-FGFR3 isolado compreendendo:(a) pelo menos um, dois, três, quatro ou cinco sequências de regiões hipervariáveis (HVR) selecionadas a partir de:(i) HVR-L1 compreendendo a sequência A1-A11, em que A1-A11 é RASQDVDTSLA (SEQ ID NO: 87);(ii) HVR-L2 compreendendo a sequência B1-B7, em que B1-B7 é SASFLYS (SEQ ID NO: 88);(iii) HVR-L3 compreendendo a sequência C1-C9, em que C1-C9 é QQSTGHPQT (SEQ ID NO: 89);(iv) HVR-H1 compreende sequência de aminoácidos D1-D10, em que D1-D10 é GFTFTSTGIS (SEQ ID NO: 84);(v) HVR-H2 compreendendo a sequência E1-E18, em que E1-E18 é GRIYPTSGSTNYADSVKG (SEQ ID NO: 85); e(vi) HVR-H3 compreendendo a sequência F1-F20, em que F1-F20 é ARTYGIYDLYVDYTEYVMDY (SEQ ID NO: 86); e (b) pelo menos uma HVR variante, onde a sequência HVR variante compreende a modificação de pelo menos um resíduo (pelo menos, dois resíduos, pelo menos três ou mais resíduos) da sequência exibida na SEQ ID Nos:1-18, 48-131 e 140-145. A modificação desejável é uma substituição, inserção ou deleção.
[031] Em alguns exemplos de realização, uma HVR-L1 variante compreende 1-6 (1, 2, 3, 4, 5 ou 6) substituições em qualquer combinação das seguintes posições: A5 (V ou D), A6 (V ou I), A7 (D, E ou S), A8 (T ou I), A9 (A ou S) e A10 (V ou L). Em alguns exemplos de realização, uma HVR-L2 variante compreende 1-2 (1 ou 2) substituições em qualquer combinação das seguintes posições: B1 (S ou G), B4 (F ou S ou T) e B6 (A ou Y). Em alguns exemplos de realização, uma HVR-L3 variante compreende 1-6 (1, 2, 3, 4, 5 ou 6)substituições em qualquer combinação das seguintes posições: C3 (G ou S ou T), C4 (T ou Y ou A), C5 (G ou S ou T ou A), C6 (A ou H ou D ou T ou N), C7 (Q ou P ou S), e C8 (S ou Y ou L ou P ou Q). Em alguns exemplos de realização, uma HVR-H2 variante compreende 1-3 (1, 2 ou 2) substituições em qualquer combinação das seguintes posições: D3 (S ou T), D5 (W ou Y ou S ou T), D6 (S ou G ou T). Em alguns exemplos de realização, uma HVR-H2 variante compreende 1-6 (1, 2, 3, 4, 5 ou 6) substituições em qualquer combinação das seguintes posições: E2 (R ou S), E6 (Y ou A ou L ou S ou T), E7 (A ou Q ou D ou G ou Y ou S ou N ou F), E8 (A ou D ou G), E9 (T ou S), E10 (K ou F ou T ou S), E11 (Y ou H ou N ou I).
[032] Em um aspecto, a presente invenção fornece um anticorpo anti-FGFR3 isolado compreendendo:(a) pelo menos um, dois, três, quatro ou cinco sequências de regiões hipervariáveis (HVR) selecionadas a partir de:(i) HVR-L1 compreendendo a sequênciaRASQX1X2X3X4X5X6A, em que X1 é V ou D, X2 é V ou I, X3 é D, E ou S, X4 é T ou I, X5 é A ou S, e X6 é V ou L (SEQ ID NO:146),(ii) HVR-L2 compreendendo a sequência X1ASFLX2S em queX1 é S ou G e X2 é A ou Y (SEQ ID NO:147),(iii) HVR-L3 compreendendo a sequência QQX1X2X3X4X5X6T, em que X1 é G, S ou T, X2 é T, Y ou A, X3 é G, S, T, ou A, X4 é A, H, D, T, ou N, X5 é Q, P ou S, X6 é S, Y, L, P ou Q (SEQ ID NO:148),(iv) HVR-H1 compreendendo a sequência GFX1FX2X3TGIS, em que X1 é S ou T, X2 é W, Y, S ou T, X3 é S, G ou T (SEQ ID NO:149),(v) HVR-H2 compreendendo a sequênciaGRIYPX1X2X3X4X5X6YADSVKG, em que X1 é Y, A, L, S, ou T, X2 é A, Q, D, G, Y, S, N ou F, X3 é A, D, ou G, X4 é T ou S, X5 é K, F, T, ou S, X6 é Y, H, N ou I (SEQ ID NO:150), e(vi) HVR-H3 compreendendo a sequência
[033] Em alguns exemplos de realização, a HVR -L1 compreende a sequência RASQX1VX2X3X4VA, em que X1 é V ou D, X2 é D, E ou S, X3 é T ou I, X4 é A ou S (SEQ ID NO: 152). Em alguns exemplos de realização, HVR-L3 compreende a sequência QQX1X2X3X4X5X6T, em que X1 é S, G, ou T, X2 é Y, T, ou A, X3 é T ou G, X4 é T, H ou N, X5 é P ou S, X6 é P, Q, Y ou L (SEQ ID NO:153). Em alguns exemplos de realização, HVR-H2 compreende a sequência GRIYPX1X2GSTX3YADSVKG, em que X1 é T ou L, X2 é N, Y, S, G, A ou Q; X3 é N ou H (SEQ ID NO:154).
[034] Em outro aspecto, a presente invenção apresenta um anticorpo anti-FGFR3 isolado que compreende um, dois, três, quatro, cinco, ou seis HVRs, em que cada HVR compreende, consiste, ou consiste essencialmente de uma sequência selecionada a partir das SEQ ID NOS:1-18, 48-131 e 140-145, e onde a SEQ ID NO: 1, 7, 13, 48, 54, 60, 66, 72, 78, 84, 90, 96, 102, 108, 114, 120, 126 ou 143 corresponde a uma HVR-H1, SEQ ID NO: 2, 8, 14, 49, 55, 61, 67, 73, 79, 85, 91, 97, 103, 109, 115, 121, 127 ou 144 corresponde a uma HVR-H2, SEQ ID NO: 3, 9, 15, 50, 56, 62, 68, 74, 80, 86, 92, 98, 104, 110, 116, 122, 128 ou 145 corresponde a uma HVR-H3, SEQ ID NO: 4, 10, 16, 51, 57, 63, 69, 75, 81, 87, 93, 99, 105, 111, 117, 123, 129 ou 140 corresponde a uma HVR-L1, SEQ ID NO: 5, 11, 17, 52, 58, 64, 70, 76, 82, 88, 94, 100, 106, 112, 118, 124, 130 ou 141 correspondem a uma HVR-L2, e SEQ ID NO: 6, 12, 18, 53, 59, 65, 71, 77, 83, 89, 95, 101, 107, 113, 119, 125, 131 ou 142 corresponde a uma HVR-L3.
[035] Em um aspecto, a presente invenção fornece um anticorpo anti-FGFR3 compreendendo uma HVR- H1 compreendendo a sequência SEQ ID NO: 1, 7, 13, 48, 54, 60, 66, 72, 78, 84, 90, 96, 102, 108, 114, 120, 126 ou 143.
[036] Em um aspecto, a presente invenção fornece um anticorpo anti-FGFR3 compreendendo uma HVR- H2 compreendendo a sequência SEQ ID NO: 2, 8, 14, 49, 55, 61, 67, 73, 79, 85, 91, 97, 103, 109, 115, 121, 127 ou 144.
[037] Em um aspecto, a presente invenção fornece um anticorpo anti-FGFR3 compreendendo uma HVR- H3 compreendendo a sequência SEQ ID NO: 3, 9, 15, 50, 56, 62, 68, 74, 80, 86, 92, 98, 104, 110, 116, 122, 128 ou 145.
[038] Em um aspecto, a presente invenção fornece um anticorpo anti-FGFR3 compreendendo uma HVR- L1 compreendendo a sequência SEQ ID NO: 4, 10, 16, 51, 57, 63, 69, 75, 81, 87, 93, 99, 105, 111, 117, 123, 129 ou 140.
[039] Em um aspecto, a presente invenção fornece um anticorpo anti-FGFR3 compreendendo uma HVR- L2 compreendendo a sequência SEQ ID NO: 5, 11, 17, 52, 58, 64, 70, 76, 82, 88, 94, 100, 106, 112, 118, 124, 130 ou 141.
[040] Em um aspecto, a presente invenção fornece um anticorpo anti-FGFR3 compreendendo uma HVR- L3 compreendendo a sequência SEQ ID NO: 6, 12, 18, 53, 59, 65, 71, 77, 83, 89, 95, 101, 107, 113, 119, 125, 131 ou 142.
[041] Em um aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo uma HVR-H1, HVR-H2, HVR- H3, em que cada uma, em ordem, compreende a SEQ ID NO: 1, 2, 3, e/ou uma região variável da cadeia leve compreendendo uma HVR-L1, HVR-L2, e HVR- L3, em que cada uma, em ordem, compreende a SEQ ID NO: 4, 5, 6.
[042] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo uma HVR-H1, HVR-H2, HVR-H3, em que cada uma, em ordem, compreende a SEQ ID NO: 7, 8, 9, e/ou uma região variável da cadeia leve compreendendo uma HVR-L1, HVR-L2, e HVR-L3, em que cada uma, em ordem, compreende a SEQ ID NO: 10, 11, 12.
[043] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo uma HVR-H1, HVR-H2, HVR-H3, em que cada uma, em ordem, compreende a SEQ ID NO: 13, 14, 15, e/ou uma região variável da cadeia leve compreendendo uma HVR-L1, HVR-L2, e HVR-L3, em que cada uma, em ordem, compreende a SEQ ID NO: 16, 17, 18.
[044] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo uma HVR-H1, HVR-H2, HVR-H3, em que cada uma, em ordem, compreende a SEQ ID NO: 48, 49, 50, e/ou uma região variável da cadeia leve HVR-L1, HVR-L2, e HVR-L3, em que cada uma, em ordem, compreende a SEQ ID NO: 51, 52, 53.
[045] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo uma HVR-H1, HVR-H2, HVR-H3, em que cada uma, em ordem, compreende a SEQ ID NO: 54, 55, 56, e/ou uma região variável da cadeia leve compreendendo uma HVR-L1, HVR-L2, e HVR-L3, em que cada uma, em ordem, compreende a SEQ ID NO: 57, 58, 59.
[046] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo uma HVR-H1, HVR-H2, HVR-H3, em que cada uma, em ordem, compreende a SEQ ID NO: 60, 61, 62, 63 e/ou uma região variável da cadeia leve compreendendo uma HVR-L1, HVR- L2, e HVR-L3, em que cada uma, em ordem, compreende a SEQ ID NO: 63, 64, 65.
[047] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo uma HVR-H1, HVR-H2, HVR-H3, em que cada uma, em ordem, compreende a SEQ ID NO: 66, 67, 68, e/ou uma região variável da cadeia leve compreendendo uma HVR-L1, HVR-L2, e HVR-L3, em que cada uma, em ordem, compreende a SEQ ID NO: 69, 70, 71.
[048] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo uma HVR-H1, HVR-H2, HVR-H3, em que cada uma, em ordem, compreende a SEQ ID NO: 72, 73, 74, e/ou uma região variável da cadeia leve compreendendo uma HVR-L1, HVR-L2, e HVR-L3, em que cada uma, em ordem, compreende a SEQ ID NO: 75, 76, 77.
[049] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo uma HVR-H1, HVR-H2, HVR-H3, em que cada uma, em ordem, compreende a SEQ ID NO: 78, 79, 80, e/ou uma região variável da cadeia leve compreendo HVR-L1, HVR-L2, e HVR- L3, em que cada uma, em ordem, compreende a SEQ ID NO: 81, 82, 83.
[050] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo uma HVR-H1, HVR-H2, HVR-H3, em que cada uma, em ordem, compreende a SEQ ID NO: 84, 85, 86, e/ou uma região variável da cadeia leve compreendendo uma HVR-L1, HVR-L2, e HVR-L3, em que cada uma, em ordem, compreende a SEQ ID NO: 87, 88, 89.
[051] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo uma HVR-H1, HVR-H2, HVR-H3, em que cada uma, em ordem, compreende a SEQ ID NO: 90, 91, 92, e/ou uma região variável da cadeia leve compreendendo uma HVR-L1, HVR-L2, e HVR-L3, em que cada uma, em ordem, compreende a SEQ ID NO: 93, 94, 95.
[052] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo uma HVR-H1, HVR-H2, HVR-H3, em que cada uma, em ordem, compreende a SEQ ID NO: 96, 97, 98, e/ou uma região variável da cadeia leve compreendendo uma HVR-L1, HVR-L2, e HVR-L3, em que cada uma, em ordem, compreende a SEQ ID NO: 99, 100, 101.
[053] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo uma HVR-H1, HVR-H2, HVR-H3, em que cada uma, em ordem, compreende a SEQ ID NO: 102, 103, 104, e/ou uma região variável da cadeia leve compreendendo uma HVR-L1, HVR-L2, e HVR-L3, em que cada uma, em ordem, compreende a SEQ ID NO: 105, 106, 107.
[054] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo uma HVR-H1, HVR-H2, HVR-H3, em que cada uma, em ordem, compreende a SEQ ID NO: 108, 109, 110, e/ou uma região variável da cadeia leve compreendendo uma HVR-L1, HVR-L2, e HVR-L3, em que cada uma, em ordem, compreende a SEQ ID NO: 111, 112, 113.
[055] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo uma HVR-H1, HVR-H2, HVR-H3, em que cada uma, em ordem, compreende a SEQ ID NO: 114, 115, 116, e/ou uma região variável da cadeia leve compreendendo uma HVR-L1, HVR-L2, e HVR-L3, em que cada uma, em ordem, compreende a SEQ ID NO: 117, 118, 119.
[056] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo uma HVR-H1, HVR-H2, HVR-H3, em que cada uma, em ordem, compreende a SEQ ID NO: 120, 121, 122, e/ou uma região variável da cadeia leve compreendendo uma HVR-L1, HVR-L2, e HVR-L3, em que cada uma, em ordem, compreende a SEQ ID NO: 123, 124, 125.
[057] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo uma HVR-H1, HVR-H2, HVR-H3, em que cada uma, em ordem, compreende a SEQ ID NO: 126, 127, 128, e/ou uma região variável da cadeia leve compreendendo uma HVR-L1, HVR-L2, e HVR-L3, em que cada uma, em ordem, compreende a SEQ ID NO: 129, 130, 131.
[058] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo uma HVR-H1, HVR-H2, HVR-H3, em que cada uma, em ordem, compreende a SEQ ID NO: 140, 141, 142, e/ou uma região variável da cadeia leve compreendendo uma HVR-L1, HVR-L2, e HVR-L3, em que cada uma, em ordem, compreende a SEQ ID NO: 143, 144, 145.
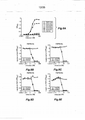
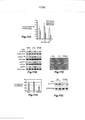
[059] As sequências de aminoácidos SEQ ID NOs: 1-18, 48-131 e 140-145 são numeradas com relação ao HVR individual (ou seja, H1, H2 e H3), como indicado na Figura 1, sendo a numeração consistente com o sistema de numeração de Kabat conforme descrito abaixo.
[060] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo a SEQ ID NO: 132 e uma região variável de cadeia leve.
[061] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia leve compreendendo a SEQ ID NO: 133 e uma região variável de cadeia pesada.
[062] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo a SEQ ID NO: 132 e uma região variável de cadeia leve compreendendo a SEQ ID NO: 133.
[063] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo a SEQ ID NO: 134 e uma região variável de cadeia leve.
[064] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia leve compreendendo a SEQ ID NO: 135 e uma região variável de cadeia pesada.
[065] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia leve compreendendo a SEQ ID NO: 139 e uma região variável de cadeia pesada.
[066] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo a SEQ ID NO: 134 e uma região variável de cadeia leve compreendendo a SEQ ID NO: 135.
[067] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo a SEQ ID NO: 136 e uma região variável de cadeia leve.
[068] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia leve compreendendo a SEQ ID NO: 137 e uma região variável de cadeia pesada.
[069] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo a SEQ ID NO: 136 e uma região variável de cadeia leve compreendendo a SEQ ID NO: 137.
[070] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo a SEQ ID NO: 138 e uma região variável de cadeia leve.
[071] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia leve compreendendo a SEQ ID NO: 139 e uma região variável de cadeia pesada.
[072] Em outro aspecto, um anticorpo anti-FGFR3 compreende uma região variável de cadeia pesada compreendendo a SEQ ID NO: 138 e uma região variável de cadeia leve compreendendo a SEQ ID NO: 139.
[073] Em um aspecto, a presente invenção fornece um anticorpoanti-FGFR3 compreendendo: pelo menos uma, duas, três, quatro cinco e/ou seissequências de regiões hipervariáveis (HVR) selecionadas a partir do grupo queconsiste em:(a) HVR-L1 compreendendo a sequência SASSSVSYMH (SEQ IDNO:155), SASSSVSYMH (SEQ ID NO:156) ou LASQTIGTWLA (SEQ IDNO:157),(b) HVR-L2 compreendendo a sequência TWIYDTSILAS (SEQ IDNO:158), RWIYDTSKLAS (SEQ ID NO:159), ou LLIYAATSLAD (SEQ IDNO:160),(c) HVR-L3 compreendendo a sequência QQWTSNPLT (SEQ IDNO:161), QQWSSYPPT (SEQ ID NO:162), ou QQLYSPPWT (SEQ ID NO:163),(d) HVR-H1 compreendendo a sequência GYSFTDYNMY (SEQ IDNO:164), GYVFTHYNMY (SEQ ID NO:165), ou GYAFTSYNMY (SEQ IDNO:166),(e) HVR-H2 compreendendo a sequênciaWIGYIEPYNGGTSYNQKFKG (SEQ ID NO:167), WIGYIEPYNGGTSYNQKFKG(SEQ ID NO:168), ou WIGYIDPYIGGTSYNQKFKG (SEQ ID NO:169), e(f) HVR-H3 compreendendo a sequência ASPNYYDSSPFAY (SEQID NO:170), ARGQGPDFDV (SEQ ID NO:171), ou ARWGDYDVGAMDY (SEQID NO:172).
[074] Em um aspecto, a presente invenção fornece um anticorpoanti-FGFR3 compreendendo: pelo menos uma, duas, três, quatro cinco e/ou seissequências de regiões hipervariáveis (HVR) selecionadas a partir do grupo queconsiste em:(a) HVR-L1 compreendendo a sequência SASSSVSYMH (SEQ IDNO:155),(b) HVR-L2 compreendendo a sequência TWIYDTSILAS (SEQ IDNO:158),(c) HVR-L3 compreendendo a sequência QQWTSNPLT (SEQ IDNO:161),(d) HVR-H1 compreendendo a sequência GYSFTDYNMY (SEQ IDNO:164),(e) HVR-H2 compreendendo a sequênciaWIGYIEPYNGGTSYNQKFKG (SEQ ID NO:167), e(f) HVR-H3 compreendendo a sequência ASPNYYDSSPFAY (SEQID NO:170).
[075] Em um aspecto, a presente invenção fornece um anticorpoanti-FGFR3 compreendendo: pelo menos uma, duas, três, quatro cinco e/ou seissequências de regiões hipervariáveis (HVR) selecionadas a partir do grupo queconsiste em:(a) HVR-L1 compreendendo a sequência SASSSVSYMH (SEQ IDNO:156),(b) HVR-L2 compreendendo a sequência RWIYDTSKLAS (SEQ IDNO:159),(c) HVR-L3 compreendendo a sequência QQWSSYPPT (SEQ IDNO:162),(d) HVR-H1 compreendendo a sequência GYVFTHYNMY (SEQ IDNO:165),(e) HVR-H2 compreendendo a sequênciaWIGYIEPYNGGTSYNQKFKG (SEQ ID NO:168), e(f) HVR-H3 compreendendo a sequência ARGQGPDFDV (SEQ IDNO:171).
[076] Em um aspecto, a presente invenção fornece um anticorpoanti-FGFR3 compreendendo: pelo menos uma, duas, três, quatro cinco e/ou seissequências de regiões hipervariáveis (HVR) selecionadas a partir do grupo queconsiste em:(a) HVR-L1 compreendendo a sequência LASQTIGTWLA (SEQ IDNO:157),(b) HVR-L2 compreendendo a sequência LLIYAATSLAD (SEQ IDNO:160),(c) HVR-L3 compreendendo a sequência QQLYSPPWT (SEQ IDNO:163),(d) HVR-H1 compreendendo a sequência GYAFTSYNMY (SEQ IDNO:166),(f) HVR-H2 compreendendo a sequênciaWIGYIDPYIGGTSYNQKFKG (SEQ ID NO:169), e(g) HVR-H3 compreendendo a sequência ARWGDYDVGAMDY(SEQ ID NO:172).
[077] Em um aspecto, a presente invenção fornece um anticorpoanti-FGFR3 compreendendo (a) uma cadeia leve compreendendo (i) HVR-L1compreendendo a sequência SASSSVSYMH (SEQ ID NO: 155), (ii) HVR-L2compreendendo a sequência TWIYDTSILAS (SEQ ID NO: 158), e (iii) HVR-L3compreendendo a sequência QQWTSNPLT (SEQ ID: 161), e/ou ( b) uma cadeiapesada compreendendo (i) HVR-H1 compreendendo a sequênciaGYSFTDYNMY (SEQ ID NO: 164), (ii) HVR-H2 compreendendo a sequênciaWIGYIEPYNGGTSYNQKFKG (SEQ ID NO: 167), e (iii) HVR-H3 compreendendoa sequência ASPNYYDSSPFAY (SEQ ID: 170).
[078] Em um aspecto, a presente invenção proporciona umPetição 870210051124, de 07/06/2021, pág. 29/51621/243anticorpo anti-FGFR3 compreendendo (a) uma cadeia leve compreendendo (i)HVR- L1 compreendendo a sequência SASSSVSYMH (SEQ ID NO: 156), (ii)HVR-L2 compreendendo a sequência RWIYDTSKLAS (SEQ ID NO: 159), e (iii)HVR-L3 compreendendo a sequência QQWSSYPPT (SEQ ID: 162), e/ou (b)uma cadeia pesada compreendendo (i) HVR- H1 compreendendo a sequênciaGYVFTHYNMY (SEQ ID NO: 165), (ii) HVR-H2 compreendendo a sequênciaWIGYIEPYNGGTSYNQKFKG (SEQ ID NO: 168), e (iii) HVR- H3compreendendo a sequência ARGQGPDFDV (SEQ ID NO: 171).
[079] Em um aspecto, a presente invenção proporciona um anticorpo anti-FGFR3 compreendendo (a) uma cadeia leve compreendendo (i) HVR-L1 compreendendo a sequência LASQTIGTWLA (SEQ ID NO:157); (ii) HVR-L2 compreendendo a sequência LLIYAATSLAD (SEQ ID NO:160); e (iii) HVR-L3 compreendendo a sequência QQLYSPPWT (SEQ ID NO:163) ; e/ou (b) uma cadeia pesada compreendendo (i) HVR-H1 compreendendo a sequência GYAFTSYNMY (SEQ ID NO:166); (ii) HVR-H2 compreendendo a sequência WIGYIDPYIGGTSYNQKFKG (SEQ ID NO:169); e (iii) HVR-H3 compreendendo a sequência ARWGDYDVGAMDY (SEQ ID NO:172). Alguns exemplos de realização de anticorpos da presente invenção compreendem um domínio variável de cadeia leve do anticorpo humanizado 4D5 (huMAb4D5-8) (Herceptin®, Genentech Inc., South San Francisco, CA, EUA) (também referido na Patente US 6.407.213 e Lee et a.l, J. Mol Biol (2004), 340 (5):1073-1093), conforme ilustrado na SEQ ID NO: 173 abaixo:1 Asp Ile Gln Met Tre Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gli Asp Arg Val Tre Ile Tre Cis Arg Ala Ser Gln Asp Val Asn Tre Ala Val Ala Trp Tir Gln Gln Lis Pro Gli Lis Ala Pro Lis Leu Leu Ile Tir Ser Ala Ser Fen Leu Tir Ser Gli Val Pro Ser Arg Fen Ser Gli Ser Arg Ser Gli Tre Asp Fen Tre Leu Tre Ile Ser Ser Leu Gln Pro Glu Asp Fen Ala Tre Tir Tir Cis Gln Gln His Tir Tre Tre Pro Pro Tre Fen Gli Gln Gli Tre Lis Val Glu Ile Lis 107 (SEQ ID NO:173) (Resíduos HVR estão sublinhados).
[080] Em um exemplo de realização, a sequência de domínio variável de cadeia leve do huMAb4D5-8 é modificada em uma ou mais das posições 30, 66 e 91 (Asn, Arg, e His, conforme indicado em negrito/itálico acima, respectivamente). Em um exemplo de realização específico, a sequência do huMAb4D5-8 modificada compreende uma Ser na posição 30, uma Gly na posição 66 e/ou uma Ser na posição 91. Assim, em uma realização, um anticorpo da presente invenção compreende um domínio variável de cadeia leve compreendendo a sequência representada na SEQ ID NO: 174, abaixo:1 Asp Ile Gln Met Tre Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gli Asp Arg Val Tre Ile Tre Cis Arg Ala Ser Gln Asp Val Ser Tre Ala Val Ala Trp Tir Gln Gln Lis Pro Gli Lis Ala Pro Lis Leu Leu Ile Tir Ser Ala Ser Fen Leu Tir Ser Gli Val Pro Ser Arg Fen Ser Gli Ser Gli Ser Gli Tre Asp Fen Tre Leu Tre Ile Ser Ser Leu Gln Pro Glu Asp Fen Ala Tre Tir Tir Cis Gln Gln Ser Tir Tre Tre Pro Pro Tre Fen Gli Gln Gli Tre Lis Val Glu Ile Lis 107 (SEQ ID NO:174) (Resíduos HVR estão sublinhados).
[081] Os resíduos substituídos com relação ao huMAb4D5-8 estão indicados em negrito/itálico.
[082] Os anticorpos da presente invenção podem compreender qualquer sequência de domínio variável da região estrutural (arcabouço ou framework) adequada, desde que a atividade de ligação ao FGFR3 seja substancialmente mantida. Por exemplo, em alguns exemplos de realização, os anticorpos da presente invenção compreendem uma sequência consenso da região estrutural de cadeia pesada subgrupo III humana. Em um exemplo de realização destes anticorpos, a sequência consenso da região estrutural compreende uma substituição na posição 71, 73, e/ou 78. Em alguns exemplos de realizações destes anticorpos, a posição 71 é A, 73 é T e/ou 78 é A. Em um exemplo de realização, estes anticorpos compreendem as sequências da região estrutural de domínio variável de cadeia pesada do huMAb4D5-8 (Herceptin ®, Genentech Inc., South San Francisco, CA, EUA) (também referido nas Patentes US 6.407.213; US 5.821.337, e em Lee et al., J. Mol. Biol. (2004), 340 (5):1073- 1093). Em um exemplo de realização, estes anticorpos compreendem ainda a sequência de consenso da região estrutural da cadeia leve KI humana. Em um exemplo de realização, estes anticorpos compreendem as sequências HVR de cadeia leve do huMAb4D5-8 conforme descrito nas Patente US 6.407.213 e US 5.821.337). Em um exemplo de realização, estes anticorpos compreendem as sequências do domínio variável de cadeia leve do huMAb4D5-8 (Herceptin ®, Genentech Inc., South San Francisco, CA, EUA) (também referido nas Patentes US 6.407.213; US 5.821.337, e em Lee et al., J. Mol. Biol. (2004), 340 (5):1073- 1093).
[083] Em uma realização, um anticorpo da presente invenção compreende um domínio variável de cadeia pesada, em que a sequência da região estrutural compreende a sequência da SEQ ID NOS:19 e 203-205, 20 e 206-208, 21 e 209-211, 22 e 212-214 , 23 e 215-217, 24 e 218-220, 25 e 221223, 26 e 224-226, 27 e 227-229, 28 e 230-232, 29 e 233-235, 30 e 236-238, 31 e 239-241, 32 e 242-244, 33 e 245-247, 34 e 248-250, 35 e 251-253, 36 e 254256, e/ou 37 e 257-259, e as sequências HVR-H1, -H2, e -H3 são SEQ ID NOS:13, 14 e/ou 15, respectivamente. Em outra realização, a sequência da região estrutural compreende a sequência da SEQ ID NOS:19 e 203-205, 20 e 206-208, 21 e 209-211, 22 e 212-214 , 23 e 215-217, 24 e 218-220, 25 e 221223, 26 e 224-226, 27 e 227-229, 28 e 230-232, 29 e 233-235, 30 e 236-238, 31 e 239-241, 32 e 242-244, 33 e 245-247, 34 e 248-250, 35 e 251-253, 36 e 254256, e/ou 37 e 257-259, e as sequências HVR-H1, -H2, e -H3 são SEQ ID NOS:48, 49 e/ou 50, respectivamente. Ainda em outra realização, a sequência da região estrutural compreende a sequência da SEQ ID NOS: 19 e 203-205, 20 e 206-208, 21 e 209-211, 22 e 212-214 , 23 e 215-217, 24 e 218-220, 25 e 221- 223, 26 e 224-226, 27 e 227-229, 28 e 230-232, 29 e 233-235, 30 e 236-238, 31 e 239-241, 32 e 242-244, 33 e 245-247, 34 e 248-250, 35 e 251-253, 36 e 254256, e/ou 37 e 257-259, e as sequências HVR-H1, -H2, e -H3 são SEQ ID NOS: 84, 85 e/ou 86, respectivamente. Em uma realização adicional, a sequência da região estrutural compreende a sequência da SEQ ID NOS: 19 e 203-205, 20 e 206-208, 21 e 209-211, 22 e 212-214 , 23 e 215-217, 24 e 218-220, 25 e 221223, 26 e 224-226, 27 e 227-229, 28 e 230-232, 29 e 233-235, 30 e 236-238, 31 e 239-241, 32 e 242-244, 33 e 245-247, 34 e 248-250, 35 e 251-253, 36 e 254256, e/ou 37 e 257-259, e as sequências HVR-H1, -H2, e -H3 são SEQ ID NOS: 108, 109 e/ou 110, respectivamente.
[084] Em um exemplo de realização específico, um anticorpo da presente invenção compreende um domínio variável de cadeia leve, no qual a sequência da região estrutural compreende a sequência SEQ ID NOS: 38 e 260262, 263-265 e 39, 40 e 266-268, e/ou 41 e 269-271, e as sequências HVR L1, L2, L3 são as SEQ ID NOS: 16, 17, e/ou 18, respectivamente. Em outro exemplo de realização, um anticorpo da presente invenção compreende um domínio variável de cadeia leve, no qual a sequência da região estrutural compreende a sequência SEQ ID NOS: 38 e 260-262, 39, 263-265 e 40 e 266-268, e/ou 41 e 269-271, e as sequências HVR L1, L2, L3 são as SEQ ID NOS: 51, 52, e/ou 53, respectivamente. Em um exemplo de realização adicional, um anticorpo da presente invenção compreende um domínio variável de cadeia leve, no qual a sequência da região estrutural compreende a sequência SEQ ID NOS: 38 e 260262, 39, 263-265 e 40 e 266-268, e/ou 41 e 269-271, e as sequências HVR L1, L2, L3 são as SEQ ID NOS: 87, 88, e/ou 89, respectivamente. Em outro exemplo de realização, um anticorpo da presente invenção compreende um domínio variável de cadeia leve, no qual a sequência da região estrutural compreende a sequência SEQ ID NOS: 38 e 260-262, 39 e 263-265, 40 e 266-268, e/ou 41 e 269-271, e as sequências HVR L1, L2, L3 são as SEQ ID NOS: 111, 112, e/ou 113, respectivamente.
[085] Em outro aspecto, um anticorpo da presente invenção compreende uma sequência de domínio variável de cadeia pesada da SEQ ID NO: 132 e/ou uma sequência de domínio variável de cadeia leve da SEQ ID NO: 133. Em outro aspecto, um anticorpo da presente invenção compreende uma sequência de domínio variável de cadeia pesada da SEQ ID NO: 134 e/ou uma sequência de domínio variável de cadeia leve da SEQ ID NO: 135. Em outro aspecto, um anticorpo da presente invenção compreende uma sequência de domínio variável de cadeia pesada da SEQ ID NO: 136 e/ou uma sequência de domínio variável de cadeia leve da SEQ ID NO: 137. Em outro aspecto, um anticorpo da presente invenção compreende uma sequência de domínio variável de cadeia pesada da SEQ ID NO: 138 e/ou uma sequência de domínio variável de cadeia leve da SEQ ID NO: 139.
[086] Em um aspecto, a presente invenção fornece um anticorpo anti-FGFR3 que se liga a um polipeptídeo compreendendo, consistindo essencialmente de, ou consistindo da seguinte sequência de aminoácidos: LAVPAANTVRFRCPA (SEQ ID NO:179) e/ou SDVEFHCKVYSDAQP (SEQ ID NO:180).
[087] Em alguns exemplos de realização, o anticorpo se liga a um polipeptídeo compreendendo, consistindo essencialmente de, ou consistindo dos aminoácidos de números 164-178 e/ou 269-283 da sequência de aminoácido do FGFR3 humano maduro.
[088] Em uma realização, um anticorpo anti-FGFR3 da presente invenção se liga especificamente uma sequência de aminoácidos que possui pelo menos 50%, 60%, 70%, 80%, 90%, 95%, 98% de identidade de sequência ou similaridade com a sequência LAVPAANTVRFRCPA (SEQ ID NO: 179) e/ou SDVEFHCKVYSDAQP (SEQ ID NO: 180).
[089] Em um aspecto, o anticorpo anti-FGFR3 da presente invenção se liga a pelo menos um, dois, três, quatro ou qualquer número, até todos os resíduos 154, 155, 158, 159, 161, 162, 163, 164, 165, 166, 167, 168, 169, 170, 171, 172, 173, 174, 175, 177, 202, 205, 207, 210, 212, 214, 216, 217, 241, 246, 247, 248, 278, 279, 280, 281, 282, 283, 314, 315, 316, 317 e/ou 318 do polipeptídeo FGFR3 IIIb, ou resíduos equivalentes do polipeptídeo FGFR3 IIIc. Um técnico hábil no assunto compreende como alinhar as sequências de FGFR3 a fim identificar os resíduos correspondentes entre sequências de FGFR3 respectivas. A combinação de dois ou mais resíduos pode incluir quaisquer resíduos 154, 155, 158, 159, 161, 162, 163, 164, 165, 166, 167, 168, 169, 170, 171, 172, 173, 174, 175, 177, 202, 205, 207, 210, 212, 214, 216, 217, 241, 246, 247, 248, 278, 279, 280, 281, 282, 283, 314, 315, 316, 317 e/ou 318 do polipeptídeo FGFR3 IIIb, ou resíduos equivalentes do polipeptídeo FGFR3 IIIc. Em alguns exemplos de realização, o anticorpo anti- FGFR3 se liga a pelo menos um, dois, três, quatro ou qualquer número, até todos os resíduos 158, 159, 169, 170, 171, 173, 175, 205, 207 e/ou 315 do polipeptídeo FGFR3 IIIb, ou resíduos equivalentes do polipeptídeo FGFR3 IIIc. Em alguns exemplos de realização, o anticorpo anti-FGFR3 se liga a pelo menos um, dois, três, quatro ou qualquer número, até todos os resíduos 158, 170, 173, 175, e/ou 315 do polipeptídeo FGFR3 IIIb, ou resíduos equivalentes do polipeptídeo FGFR3 IIIc.
[090] Em um aspecto, a presente invenção fornece um anticorpo anti-FGFR3 que compete com qualquer um dos anticorpos mencionados acima para se ligar ao FGFR3. Em um aspecto, a presente invenção fornece um anticorpo anti-FGFR3 que se liga ao mesmo epítopo ou a um epítopo similar sobre o FGFR3 tal como qualquer um dos anticorpos mencionados acima.
[091] Tal como é conhecido no estado da técnica, e como descrito em maior detalhe abaixo no presente, a posição/limite do aminoácido que delineia uma região hipervariável de um anticorpo pode variar, dependendo do contexto e das várias definições conhecidas no estado da técnica (como descrito abaixo). Algumas posições dentro de um domínio variável podem ser consideradas como posições hipervariáveis híbridas em que estas posições podem ser consideradas dentro de uma região hipervariável sob um conjunto de critérios sendo que ao mesmo tempo em que se considera fora de uma região hipervariável sob um conjunto de critérios diferentes. Uma ou mais dessas posições também podem ser encontradas em regiões hipervariáveis estendidas (conforme definido mais adiante).
[092] Em algumas realizações, o anticorpo é um anticorpo monoclonal. Em outras realizações, o anticorpo é um anticorpo policlonal. Em algumas realizações, o anticorpo é selecionado a partir do grupo que consiste em um anticorpo quimérico, anticorpo maturado por afinidade, anticorpo humanizado, e um anticorpo humano. Em certas realizações, o anticorpo é um fragmento de anticorpo. Em algumas realizações, o anticorpo é um Fab, Fab', Fab'-SH, F(ab')2, ou scFv.
[093] Em alguns exemplos de realização, o anticorpo FGFR3 é um anticorpo de um braço (ou seja, o domínio variável de cadeia pesada e o domínio variável de cadeia leve formam um único braço de ligação com antígeno) compreendendo uma região Fc, em que a região Fc compreende um primeiro e um segundo polipeptídeo Fc, onde o primeiro e segundo polipeptídeo Fc estão presentes em um complexo e formam uma região Fc, que aumenta a estabilidade do referido fragmento de anticorpo quando comparado com uma molécula Fab compreendendo o dito braço de ligação ao antígeno. Vide, por exemplo, a publicação WO2006/015371.
[094] Em um exemplo de realização, o anticorpo é um anticorpo quimérico, por exemplo, um anticorpo compreendendo as sequências de ligação ao antígeno a partir de um doador não humano, enxertadas em uma sequência heteróloga não humana, humana ou humanizada (por exemplo, sequências da região estrutural e/ou do domínio constante). Em um exemplo de realização, o dito doador não humano é um camundongo. Em um exemplo de realização adicional, uma sequência de ligação ao antígeno é sintética, por exemplo, obtida por mutagênese (por exemplo, seleção por exibição de fago, etc.). Em uma realização específica, um anticorpo quimérico da presente invenção tem regiões V murina e região C humana. Em um exemplo de realização, a região V de cadeia leve murina está fundida a uma cadeia leve kappa humana. Em outro exemplo de realização, a região V de cadeia pesada murina está fundida a uma região C da IgG1 humana.
[095] Anticorpos humanizados da presente invenção incluem aqueles que têm substituições de aminoácidos na região estrutural humana (ou de arcabouço - framework )(FR) e variantes de maturação por afinidade com alterações nos CDRs enxertados. Os aminoácidos substituídos no CDR ou FR não são limitados àqueles presentes no anticorpo doador ou receptor. Em outros exemplos de realização, os anticorpos da presente invenção compreendem adicionalmente alterações nos resíduos de aminoácidos na região Fc que levam a uma função efetora melhorada incluindo uma melhor função CDC e/ou ADCC e de destruição de células B. Outros anticorpos da presente invenção incluem aqueles que possuem alterações específicas que melhoram a estabilidade. Em outros exemplos de realização, os anticorpos da presente invenção compreendem alterações nos resíduos de aminoácidos na região Fc que levam a uma diminuição da função efetora, por exemplo, diminuição da função CDC e/ou ADCC e diminuição da destruição de células B. Em alguns exemplos de realização, os anticorpos da presente invenção são caracterizados pela diminuição de ligação (tal como a ausência de ligação) ao fator C1q do sistema complemento humano e/ou receptor Fc nas células Matadoras Naturais (natural killer - NK) humanas. Em alguns exemplos de realização, os anticorpos da presente invenção são caracterizados pela diminuição de ligação (tal como a ausência de ligação) ao FCYRI, FCYRIIA, e/ou FCYRIIIA humano. Em alguns exemplos de realização, os anticorpos da presente invenção são da classe IgG (por exemplo, IgG1 ou IgG4) e inclui pelo menos uma mutação no E233, L234, G236, D265, D270, N297, E318, K320, K322, A327, A330, P331 e/ou P329 (numeração de acordo com o índice EU). Em alguns exemplos de realização, os anticorpos compreendem as mutações L234A/L235A ou D265A/N297A.
[096] Quando o anticorpo compreende uma região Fc, os carboidratos ligados a estes podem ser alterados. Por exemplo, anticorpos com uma estrutura de carboidrato madura que carece de fucose anexada a uma região Fc do anticorpo são descritos no Pedido de Patente US 2003/0157108 (Presta, L.). Veja também a US 2004/0093621 (Kyowa Hakko Kogyo Co., Ltd). Anticorpos com uma bissecção por N-acetilglicosamina (GlcNAc) nos carboidratos ligados a uma região Fc do anticorpo são referidos na publicação WO 2003/011878, Jean-Mairet et al. e na Patente US 6.602.684, Humana et al. Anticorpos com pelo menos um resíduo de galactose no oligossacarídeo ligado a uma região Fc do anticorpo são descritos na publicação WO 1997/30087, Patel et al. Veja, também, a publicação WO 1998/58964 (Raju, S.) e WO 1999/22764 (Raju, S.) sobre anticorpos com carboidratos alterados anexados à região Fc deste. Vide também US 2005/0123546 (Umana et al.) sobre moléculas de ligação a antígenos com glicosilação modificada. Em um aspecto, a presente invenção fornece polipeptídeos de ligação ao FGFR3 compreendendo qualquer uma das sequências de ligação ao antígeno contida no presente documento, em que os polipeptídeos de ligação ao FGFR3 se ligam especificamente a um FGFR3, por exemplo, um FGFR3 humano e/ou de cinomologo e/ou de camundongo.
[097] Os anticorpos da presente invenção se ligam (tal como uma ligação específica) ao FGFR3 (por exemplo, FGFR3-IIIb e/ou FGFR3-IIIc), e em alguns exemplos de realização, podem modular (por exemplo, inibir) um ou mais aspectos de sinalização do FGFR3 (tal como a fosforilação de FGFR3) e/ou interromper qualquer via biologicamente ativa relevante do FGFR3 e/ou do ligante de FGFR3, e/ou podem tratar e/ou prevenir um tumor, distúrbio proliferativo celular ou câncer, e/ou tratar e/ou prevenir uma doença associada com a expressão de FGFR3/ ou atividade de FGFR3 (tal como uma expressão e/ou atividade de FGFR3 aumentada). Em alguns exemplos de realização, o anticorpo FGFR3 se liga especificamente a um polipeptídeo consistindo ou que consiste essencialmente de um FGFR3 (por exemplo, um FGFR3 humano ou de camundongo). Em alguns exemplos de realização, o anticorpo se liga especificamente ao FGFR3 com uma Kd de 1 x 10-7 M ou mais forte.
[098] Em alguns exemplos de realização, o anticorpo anti-FGFR3 da presente invenção não é um anticorpo anti-FGFR3 descrito na patente US 2005/0147612 (por exemplo, anticorpos MSPRO2, MSPRO12, MSPRO59, MSPRO11, MSPRO21, MSPRO24, MSPRO26, MSPRO28, MSPRO29, MSPRO43, MSPRO55), anticorpo descrito em Rauchenberger et al., J Biol Chem 278 (40):38194-38205 (2003); anticorpo descrito na publicação PCTWO2006/048877 (por exemplo, anticorpos PRO-001), anticorpo descrito em Martinez Torrecuadrada et al., Mol Cancer There (2008) 7(4):862-873 (por exemplo, scFvαFGFR3 3C), anticorpo descrito em Direnzo, R et al. (2007) Proceedings of AACR Annual Meeting. Resumo 2080 (por exemplo, D11), ou um anticorpo descrito no WO 2010/002862 (por exemplo, anticorpos 15D8, 27H2, 4E7, 2G4, 20B4). Em um aspecto, a presente invenção fornece composições que compreendem um ou mais anticorpos da presente invenção e um veículo. Em uma realização, o veículo é farmaceuticamente aceitável.
[099] Em outro aspecto, a presente invenção fornece ácidos nucleicos codificando um anticorpo FGFR3 da presente invenção.
[100] Em um aspecto adicional, a presente invenção fornece vetores contendo o ácido nucleico da presente invenção.
[101] Em outro aspecto, a presente invenção fornece composições que compreendem um ou mais ácidos nucleicos da presente invenção e um veículo. Em uma realização, o veículo é farmaceuticamente aceitável.
[102] Em um aspecto, a presente invenção fornece células hospedeiras compreendendo um ácido nucleico ou um vetor da presente invenção. Um vetor pode ser de qualquer tipo, por exemplo, um vetor recombinante, tal como um vetor de expressão. Pode ser utilizada qualquer uma de uma variedade de células hospedeiras. Em uma realização, uma célula hospedeira é uma célula procariótica, por exemplo, E. coli. Em outra realização, uma célula hospedeira é uma célula eucariota, por exemplo, uma célula de mamíferos, tal como células de ovário de hamster chinês (CHO).
[103] Em outro aspecto, a presente invenção fornece métodos para produção de um anticorpo da presente invenção. Por exemplo, a presente invenção fornece métodos de produção de um anticorpo anti-FGFR3 (que, tal como definido no presente, inclui um anticorpo de comprimento total e fragmentos dos mesmos), compreendendo o dito método a expressão de um vetor recombinante da presente invenção que codifica o anticorpo em uma célula hospedeira adequada, e recuperação do anticorpo. Em alguns exemplos de realização, o método compreende o cultivo de uma célula hospedeira que compreende o ácido nucleico que codifica o anticorpo de modo que o ácido nucleico seja expresso. Em alguns exemplos de realização, o método compreende ainda a recuperação do anticorpo a partir da cultura de célula hospedeira. Em alguns exemplos de realização, o anticorpo é recuperado a partir do meio de cultura da célula hospedeira. Em alguns exemplos de realização, o método compreende ainda a combinação do anticorpo recuperado com um veículo farmaceuticamente aceitável, excipiente, ou transportador para preparar uma formulação farmacêutica compreendendo o anticorpo humanizado.
[104] Em um aspecto, a presente invenção fornece um artigo de manufatura compreendendo um recipiente; e uma composição contida dentro do recipiente, no qual a composição compreende um ou mais anticorpos FGFR3 da presente invenção. Em uma realização, a composição compreende um ácido nucleico da presente invenção. Em outra realização, uma composição compreendendo um anticorpo que contém ainda um veículo, que em algumas realizações é farmaceuticamente aceitável. Em um exemplo de realização, um artigo manufaturado da presente invenção compreende de instruções para a administração da composição (por exemplo, o anticorpo) a um indivíduo (tal como, instruções para qualquer um dos métodos descritos no presente).
[105] Em um aspecto, a presente invenção fornece um artigo de manufatura compreendido por um recipiente e uma composição contida dentro do recipiente, no qual a composição contém um ou mais anticorpos anti-FGFR3 da presente invenção; e um segundo recipiente contendo um tampão. Em uma realização, o tampão é farmaceuticamente aceitável. Em uma realização, uma composição compreende um anticorpo que consta adicionalmente de um veículo, que em alguns exemplos de realização é farmaceuticamente aceitável. Em outro exemplo de realização, um kit compreende ainda instruções para a administração da composição (por exemplo, o anticorpo) a um indivíduo.
[106] Em outro aspecto, a presente invenção fornece um anticorpo anti-FGFR3 da presente invenção para uso como um medicamento.
[107] Em outro aspecto, a presente invenção fornece um anticorpo anti-FGFR3 da presente invenção para uso no tratamento ou prevenção de uma doença, tal como uma condição patológica associada com a ativação e/ou expressão de FGFR3 (e em algumas realizações, uma superexpressão). Em alguns exemplos de realização, o distúrbio é um câncer, tumor e/ou distúrbio da proliferação celular. Em alguns exemplos de realização, o câncer, tumor e/ou distúrbio da proliferação celular é o mieloma múltiplo ou câncer de bexiga (por exemplo, carcinoma de células de transição), câncer de mama ou câncer de fígado.
[108] Em outro aspecto, a presente invenção fornece um anticorpo anti-FGFR3 da presente invenção para uso no tratamento ou prevenção de uma doença tal como um distúrbio do sistema esquelético. Em alguns exemplos de realização, o distúrbio é a acondroplasia, hipocondroplasia, nanismo, displasia tanatofórica (TD, formas clínicas TD1 e TDII), ou síndrome de craniosinostose.
[109] Em outro aspecto, a presente invenção fornece um anticorpo anti-FGFR3 da presente invenção para uso na redução da proliferação celular.
[110] Em um aspecto adicional, a presente invenção fornece um anticorpo anti-FGFR3 da presente invenção para uso na destruição de células. Em algumas realizações, a célula é uma célula de mieloma múltiplo. Em algumas realizações, a destruição da célula ocorre pela ADCC. Em algumas realizações, o anticorpo é um anticorpo nu. Em alguns exemplos de realização, a célula superexpressa FGFR3.
[111] Em um aspecto adicional, a presente invenção fornece um anticorpo anti-FGFR3 da presente invenção para uso na depleção de células, tal como células de mieloma múltiplo. Em algumas realizações, a destruição da célula ocorre pela ADCC. Em algumas realizações, o anticorpo é um anticorpo nu. Em alguns exemplos de realização, a célula superexpressa FGFR3.
[112] Em um aspecto adicional, a presente invenção fornece o uso do anticorpo anti-FGFR3 da presente invenção na preparação de um medicamento para o tratamento terapêutico e/ou profilático de um distúrbio, tal como uma condição patológica associada com a ativação e/ou expressão de FGFR3 (em alguns exemplos de realização, uma superexpressão). Em alguns exemplos de realização, o distúrbio é um câncer, tumor e/ou distúrbio da proliferação celular. Em alguns exemplos de realização, o câncer, tumor e/ou distúrbio da proliferação celular é o mieloma múltiplo ou câncer de bexiga (por exemplo, carcinoma de células de transição), câncer de mama ou câncer de fígado. Em alguns exemplos de realização, o distúrbio é um distúrbio do sistema esquelético, por exemplo, acondroplasia, hipocondroplasia, nanismo, displasia tanatofórica (TD, formas clínicas TD1 e TDII), ou síndrome de craniosinostose.
[113] Em um aspecto, a presente invenção fornece o uso de um ácido nucleico da presente invenção na preparação de um medicamento para o tratamento terapêutico e/ou profilático de um distúrbio, tal como uma condição patológica associada com a ativação e/ou expressão de FGFR3 (em alguns exemplos de realização, uma superexpressão). Em alguns exemplos de realização, o distúrbio é um câncer, tumor e/ou distúrbio da proliferação celular. Em alguns exemplos de realização, o câncer, tumor e/ou distúrbio da proliferação celular é o mieloma múltiplo ou câncer de bexiga (por exemplo, carcinoma de células de transição), câncer de mama ou câncer de fígado. Em alguns exemplos de realização, o distúrbio é um distúrbio do sistema esquelético, por exemplo, acondroplasia, hipocondroplasia, nanismo, displasia tanatofórica (TD, formas clínicas TD1 e TDII), ou síndrome de craniosinostose.
[114] Em outro aspecto, a presente invenção fornece o uso de um vetor de expressão da presente invenção na preparação de um medicamento para o tratamento terapêutico e/ou profilático de um distúrbio, tal como uma condição patológica associada com a ativação e/ou expressão de FGFR3 (em alguns exemplos de realização, uma superexpressão). Em alguns exemplos de realização, o distúrbio é um câncer, tumor e/ou distúrbio da proliferação celular. Em alguns exemplos de realização, o câncer, tumor e/ou distúrbio da proliferação celular é o mieloma múltiplo ou câncer de bexiga (por exemplo, carcinoma de células de transição), câncer de mama ou câncer de fígado. Em alguns exemplos de realização, o distúrbio é um distúrbio do sistema esquelético, por exemplo, acondroplasia, hipocondroplasia, nanismo, displasia tanatofórica (TD, formas clínicas TD1 e TDII), ou síndrome de craniosinostose.
[115] Ainda em outro aspecto adicional, a presente invenção fornece o uso de uma célula hospedeira da presente invenção na preparação de um medicamento para o tratamento terapêutico e/ou profilático de um distúrbio, tal como uma condição patológica associada com a ativação e/ou expressão de FGFR3 (em alguns exemplos de realização, uma superexpressão). Em alguns exemplos de realização, o distúrbio é um câncer, tumor e/ou distúrbio da proliferação celular. Em alguns exemplos de realização, o câncer, tumor e/ou distúrbio da proliferação celular é o mieloma múltiplo ou câncer de bexiga (por exemplo, carcinoma de células de transição), câncer de mama ou câncer de fígado. Em alguns exemplos de realização, o distúrbio é um distúrbio do sistema esquelético, por exemplo, acondroplasia, hipocondroplasia, nanismo, displasia tanatofórica (TD, formas clínicas TD1 e TDII), ou síndrome de craniosinostose.
[116] Em um aspecto adicional, a presente invenção fornece o uso de um artigo manufaturado da presente invenção na preparação de um medicamento para o tratamento terapêutico e/ou profilático de um distúrbio, tal como uma condição patológica associada com a ativação e/ou expressão de FGFR3 (em alguns exemplos de realização, uma superexpressão). Em alguns exemplos de realização, o distúrbio é um câncer, tumor e/ou distúrbio da proliferação celular. Em alguns exemplos de realização, o câncer, tumor e/ou distúrbio da proliferação celular é o mieloma múltiplo ou câncer de bexiga (por exemplo, carcinoma de células de transição), câncer de mama ou câncer de fígado. Em alguns exemplos de realização, o distúrbio é um distúrbio do sistema esquelético, por exemplo, acondroplasia, hipocondroplasia, nanismo, displasia tanatofórica (TD, formas clínicas TD1 e TDII), ou síndrome de craniosinostose.
[117] Em um aspecto, a presente invenção fornece também o uso de um kit da presente invenção na preparação de um medicamento para o tratamento terapêutico e/ou profilático de um distúrbio, tal como uma condição patológica associada com a ativação e/ou expressão de FGFR3 (em alguns exemplos de realização, uma superexpressão). Em alguns exemplos de realização, o distúrbio é um câncer, tumor e/ou distúrbio da proliferação celular. Em alguns exemplos de realização, o câncer, tumor e/ou distúrbio da proliferação celular é o mieloma múltiplo ou câncer de bexiga (por exemplo, carcinoma de células de transição), câncer de mama ou câncer de fígado. Em alguns exemplos de realização, o distúrbio é um distúrbio do sistema esquelético, por exemplo, acondroplasia, hipocondroplasia, nanismo, displasia tanatofórica (TD, formas clínicas TD1 e TDII), ou síndrome de craniosinostose.
[118] Em outro aspecto, a presente invenção fornece o uso de um anticorpo anti-FGFR3 da presente invenção para a preparação de um medicamento para a inibição da proliferação celular. Em um aspecto adicional, a presente invenção fornece o uso de um anticorpo anti-FGFR3 da presente invenção na preparação de um medicamento para matar células. Em algumas realizações, a célula é uma célula de mieloma múltiplo. Em algumas realizações, a destruição da célula ocorre pela ADCC. Em algumas realizações, o anticorpo é um anticorpo nu. Em alguns exemplos de realização, a célula superexpressa FGFR3.
[119] Em um aspecto adicional, a presente invenção fornece o uso de um anticorpo anti-FGFR3 da presente invenção na preparação de um medicamento para a depleção de células, tal como células de mieloma múltiplo. Em algumas realizações, a destruição da célula ocorre pela ADCC. Em algumas realizações, o anticorpo é um anticorpo nu. Em alguns exemplos de realização, a célula superexpressa FGFR3.
[120] A presente invenção fornece métodos e composições úteis para modular distúrbios associados com a expressão e/ou sinalização de FGFR3, tal como a expressão e/ou sinalização aumentada e/ou a expressão e/ou sinalização indesejada.
[121] Métodos da presente invenção podem ser usados para afetar qualquer estado patológico adequado. Distúrbios exemplares são descritos no presente e incluem cânceres selecionados a partir do grupo constituído por câncer de pulmão de células não pequenas, câncer de ovário, câncer de tireoide, câncer testicular, câncer endometrial, câncer de cabeça e pescoço, câncer de cérebro (por exemplo, neuroblastoma ou meningioma), câncer de pele (por exemplo, melanoma, carcinoma basocelular ou carcinoma de células escamosas) câncer da bexiga (por exemplo, carcinoma de células de transição), carcinoma da mama, câncer gástrico, câncer colorretal (CRC), carcinoma hepatocelular, câncer cervical, câncer de pulmão, câncer pancreático, câncer de próstata, e doenças hematológicas malignas (por exemplo, leucemia aguda linfoblástica de células T (LLA-T), leucemia linfoblástica aguda de células B (LLA-B), leucemia mieloide aguda (LMA), células B malignas, linfoma de Hodgkin e mieloma múltiplo). Em alguns exemplos de realização, o distúrbio é o carcinoma de células de transição invasivo. Em algumas realizações, o distúrbio é mieloma múltiplo. Exemplos de distúrbios adicionais incluem distúrbio do sistema esquelético, tal como acondroplasia, hipocondroplasia, nanismo, displasia tanatofórica (TD, formas clínicas TD1 e TDII), ou síndrome de craniosinostose.
[122] Em alguns exemplos de realização, o câncer expressa FGFR3, FGFR3 amplificado, FGFR3 translocado, e/ou FGFR3 mutante. Em certos exemplos de realização, o câncer expressa EGFR3 ativado. Em alguns exemplos de realização, o câncer expressa FGFR3 translocado (por exemplo, uma translocação t(4, 14)). Em certos exemplos de realização, o câncerexpressa EGFR3 constitutivo. Em alguns exemplos de realização, o FGFR3 constitutivo compreende uma mutação no domínio da tirosina quinase e/ou domínio justamembrana e/ou um domínio de ligação ao ligante. Em certos exemplos de realização, o câncer expressa EGFR3 independente de ligante. Em certos exemplos de realização, o câncer expressa EGFR3 dependente de ligante.
[123] Em alguns exemplos de realização, o câncer expressa FGFR3 compreendendo uma mutação correspondente a FGFR3-IIIbS248C. Em alguns exemplos de realização, o câncer expressa FGFR3-IIIbS248C e/ou FGFR3- IIIcS248C.
[124] Em alguns exemplos de realização, o câncer expressa FGFR3 compreendendo uma mutação correspondente a FGFR3-IIIbK652E. Em alguns exemplos de realização, o câncer expressa FGFR3-IIIbK652E e/ou FGFR3- IIIcK650E.
[125] Em que o FGFR3 compreende uma mutação correspondente a FGFR3-IIIbS249C. Em alguns exemplos de realização, o câncer expressa FGFR3-IIIbS249C e/ou FGFR3-IIIcS249C.
[126] Em alguns exemplos de realização, o câncer expressa FGFR3 compreendendo uma mutação correspondente a FGFR3-IIIbG372C. Em alguns exemplos de realização, o câncer expressa FGFR3-IIIbG372C e/ou FGFR3- IIIcG370C.
[127] Em alguns exemplos de realização, o câncer expressa FGFR3 compreendendo uma mutação correspondente a FGFR3-IIIbY375C. Em alguns exemplos de realização, o câncer expressa FGFR3-IIIbY375C e/ou FGFR3- IIIcY373C.
[128] Em alguns exemplos de realização, o câncer expressa (a) FGFR3-IIIbK652E e (b) um ou mais dos FGFR3-IIIbR248C, FGFR3-IIIbY375C, FGFR3- IIIbS249C e FGFR3-IIIbG372C.
[129] Em alguns exemplos de realização, o câncer expressa (a) FGFR3-IIIbR248C e (b) um ou mais dos FGFR3-IIIbK652E, FGFR3-IIIbY375C, FGFR3- IIIbS249C e FGFR3-IIIbG372C.
[130] Em alguns exemplos de realização, o câncer expressa (a) FGFR3-IIIbG372C e (b) um ou mais dos FGFR3-IIIbK652E, FGFR3-IIIbY375C, FGFR3- IIIbS249C e FGFR3-IIIbR248C.
[131] Em alguns exemplos de realização, o câncer expressa FGFR3-IIIbR248C, FGFR3-IIIbK652E, FGFR3-IIIbY375C, FGFR3-IIIbS249C, e FGFR3- IIIbG372C.
[132] Em alguns exemplos de realização, o câncer expressa níveis aumentos de fosfo-FGFR3, fosfo-FRS2 e/ou fosfo-MAPK em relação a uma amostra controle (por exemplo, uma amostra de tecido normal) ou nível controle.
[133] Em alguns exemplos de realização, o câncer expressa (por exemplo, na superfície da célula) cerca de 10.000 moléculas FGFR3 por célula ou mais (tal como 11.000, 12.000, 13.000, 14.000, 15.000, 16.000, 17.000, 18.000 ou mais receptores FGFR3). Em alguns exemplos de realização, o câncer expressa cerca de 13.000 moléculas de EGFR3. Em outros exemplos de realização, o câncer expressa cerca de 5.000, 6.000, 7.000, 8.000, ou mais moléculas FGFR3. Em alguns exemplos de realização, o câncer expressa menos que cerca de 4.000, 3.000, 2.000, 1.000, ou menos moléculas FGFR3. Em alguns exemplos de realização, o câncer expressa menos que cerca de 1000 moléculas de EGFR3.
[134] Em um exemplo de realização, uma célula que é alvo em um método da presente invenção é uma célula cancerosa. Por exemplo, uma célula de câncer pode ser uma selecionada a partir do grupo que consiste em uma célula de câncer de mama, uma célula de câncer colo retal, uma célula de câncer de pulmão (por exemplo, célula câncer de pulmão tipo células não pequenas), uma célula de câncer de tireoide, uma célula de mieloma múltiplo, uma célula de câncer testicular, uma célula de carcinoma papilar, uma célula de câncer de cólon, uma célula de câncer de pâncreas, uma célula de câncer de ovário, uma célula de câncer cervical, uma célula de câncer do sistema nervoso central, uma célula de sarcoma osteogênico, uma célula de carcinoma renal, uma célula de carcinoma hepatocelular, uma célula de câncer de bexiga (por exemplo, uma célula de carcinoma de células de transição), uma célula de carcinoma gástrico, uma célula de câncer de cabeça e pescoço, uma célula de carcinoma escamoso, uma célula de melanoma, uma célula de leucemia, uma célula de mieloma múltiplo (por exemplo, uma célula de mieloma múltiplo compreendendo em uma translocação t(4:14) no FGFR3) e uma célula de adenoma de cólon. Em um exemplo de realização, uma célula que é alvo em um método da presente invenção é uma célula hiperproliferativa e/ou hiperplásica. Em outro exemplo de realização, uma célula que é alvo em um método da presente invenção é uma célula displásica. Em outro exemplo de realização, uma célula que é alvo em um método da presente invenção é uma célula metastática.
[135] Em um aspecto, a presente invenção fornece métodos para inibir a proliferação celular em um paciente, compreendendo o dito o método em administrar ao paciente uma quantidade eficaz de um anticorpo anti-FGFR3 para reduzir a proliferação celular.
[136] Em um aspecto, a presente invenção fornece métodos para matar uma célula em um paciente, compreendendo o dito o método em administrar ao paciente uma quantidade eficaz de um anticorpo anti-FGFR3 para matar uma célula. Em algumas realizações, a célula é uma célula de mieloma múltiplo. Em algumas realizações, a destruição da célula ocorre pela ADCC. Em algumas realizações, o anticorpo é um anticorpo nu. Em alguns exemplos de realização, a célula superexpressa FGFR3.
[137] Em um aspecto, a presente invenção fornece métodos para depleção de células em um paciente, compreendendo o dito o método em administrar ao paciente uma quantidade eficaz de um anticorpo anti-FGFR3 para matar uma célula. Em algumas realizações, a destruição da célula ocorre pela ADCC. Em algumas realizações, o anticorpo é um anticorpo nu. Em alguns exemplos de realização, a célula superexpressa FGFR3.
[138] Em um aspecto, a presente invenção fornece métodos para tratar ou prevenir um distúrbio no esqueleto. Em alguns exemplos de realização, o distúrbio é a acondroplasia, hipocondroplasia, nanismo, displasia tanatofórica (TD, formas clínicas TD1 e TDII), ou síndrome de craniosinostose.
[139] Métodos da presente invenção podem ainda compreender etapas adicionais de tratamento. Por exemplo, em uma realização, um método inclui ainda uma etapa em que uma célula alvo e/ou tecido (por exemplo, uma célula cancerosa) é exposta ao tratamento de radiação ou a um agente quimioterápico.
[140] Em um aspecto, a presente invenção fornece métodos que compreendem a administração de uma quantidade eficaz de um anticorpo anti- FGFR3 em combinação com uma quantidade eficaz de um agente terapêutico (tal como um agente anti-angiogênese, outro anticorpo, um agente quimioterápico, um agente citotóxico, um agente imunossupressor, uma pró- droga, uma citocina, radioterapia citotóxica, um corticosteroide, um antiemético, uma vacina contra o câncer, um analgésico, ou um agente inibidor do crescimento). Por exemplo, os anticorpos anti-FGFR3 são usados em combinação com um agente anticâncer ou um agente anti-angiogênico para tratar diversas condições neoplásicas ou não-neoplásicas. Em exemplos específicos, os anticorpos anti-FGFR3 são usados em combinação com velcade, revlimide, tamoxifeno, letrozol, exemestano, anastrozol, irinotecan, cetuximab, fulvestrant, vinorelbina, bevacizumab, vincristina, cisplatina, vinblastina gemcitabina, metotrexato, carboplatina, paclitaxel, docetaxel, pemetrexado, 5- fluorouracil, doxorrubicina, bortezomib, lenalidomida, dexametasona, melfalina, prednisona, vincristina e/ou talidomida.
[141] Dependendo da indicação do câncer específico a ser tratado, a terapia de combinação da presente invenção pode ser combinada com agentes terapêuticos adicionais, tais como os quimioterápicos, ou terapias complementares, como a radioterapia ou cirurgia. Muitos agentes quimioterápicos conhecidos podem ser utilizados na terapia de combinação da presente invenção. Preferencialmente os agentes quimioterápicos que são padrões para o tratamento de indicações específicas serão utilizados. A dosagem ou frequência de cada agente terapêutico que será utilizado na combinação é, preferencialmente, a mesma ou menor que, a dosagem ou frequência do agente correspondente quando utilizado sem o(s) outro(s) agente(s).
[142] Em outro aspecto, a presente invenção fornece qualquer um dos anticorpos anti-FGFR3 descritos no presente, de modo que o anticorpo anti- FGFR3 compreende um marcador detectável.
[143] Em outro aspecto, a presente invenção fornece um complexo de qualquer um dos anticorpos anti-FGFR3 descrito no presente e FGFR3. Em alguns exemplos de realização, o complexo é in vivo ou in vitro. Em algumas realizações, o complexo compreende uma célula de câncer. Em algumas realizações, o anticorpo anti-FGFR3 é marcado para a detecção.BREVE DESCRIÇÃO DAS FIGURAS
[144] Figuras 1A, 1B e 1C: Sequências da alça HVR de cadeia pesada e cadeia leve de anticorpos anti-FGFR3. As figuras exibem as sequências HVR da cadeia pesada, H1, H2 e H3, e as sequências HVR da cadeia leve, L1, L2 e L3. A numeração da sequência são as seguintes:Clone 184.6 (HVR-H1 é a SEQ ID NO: 1; HVR-H2 é a SEQ ID NO: 2; HVR-H3 é a SEQ ID NO: 3; HVR-L1 é a SEQ ID NO: 4; HVR-L2 é a SEQ ID NO: 5; HVR-L3 é a SEQ ID NO: 6);Clone 184.6.1 (HVR-H1 é a SEQ ID NO: 7; HVR-H2 é a SEQ ID NO: 8; HVR-H3 é a SEQ ID NO: 9; HVR-L1 é a SEQ ID NO: 10; HVR-L2 é a SEQ ID NO: 11; HVR-L3 é a SEQ ID NO: 12);Clone 184.6.58 (HVR-H1 é a SEQ ID NO: 13; HVR-H2 é a SEQ ID NO: 14; HVR-H3 é a SEQ ID NO: 15; HVR-L1 é a SEQ ID NO: 16; HVR-L2 é a SEQ ID NO: 17; HVR-L3 é a SEQ ID NO: 18); Clone 184.6.62 (HVR-H1 é a SEQ ID NO: 48; HVR-H2 é a SEQ ID NO: 49; HVR-H3 é a SEQ ID NO: 50; HVR-L1 é a SEQ ID NO: 51; HVR-L2 é aSEQ ID NO: 52; HVR-L3 é a SEQ ID NO: 53);Clone 184.6.21 (HVR-H1 é a SEQ ID NO: 54; HVR-H2 é a SEQ ID NO: 55; HVR-H3 é a SEQ ID NO: 56; HVR-L1 é a SEQ ID NO: 57; HVR-L2 é aSEQ ID NO: 58; HVR-L3 é a SEQ ID NO: 59);Clone 184.6.49 (HVR-H1 é a SEQ ID NO: 60; HVR-H2 é a SEQ ID NO: 61; HVR-H3 é a SEQ ID NO: 62; HVR-L1 é a SEQ ID NO: 63; HVR-L2 é aSEQ ID NO: 64; HVR-L3 é a SEQ ID NO: 65);Clone 184.6.51 (HVR-H1 é a SEQ ID NO: 66; HVR-H2 é a SEQ ID NO: 67; HVR-H3 é a SEQ ID NO: 68; HVR-L1 é a SEQ ID NO: 69; HVR-L2 é aSEQ ID NO: 70; HVR-L3 é a SEQ ID NO: 71);Clone 184.6.52 (HVR-H1 é a SEQ ID NO: 72; HVR-H2 é a SEQ ID NO: 73; HVR-H3 é a SEQ ID NO: 74; HVR-L1 é a SEQ ID NO: 75; HVR-L2 é aSEQ ID NO: 76; HVR-L3 é a SEQ ID NO: 77);Clone 184.6.92 (HVR-H1 é a SEQ ID NO: 78; HVR-H2 é a SEQ ID NO: 79; HVR-H3 é a SEQ ID NO: 80; HVR-L1 é a SEQ ID NO: 81; HVR-L2 é aSEQ ID NO: 82; HVR-L3 é a SEQ ID NO: 83);Clone 184.6.1.N54S (HVR-H1 é a SEQ ID NO: 84; HVR-H2 é aSEQ ID NO: 85; HVR-H3 é a SEQ ID NO: 86; HVR-L1 é a SEQ ID NO: 87; HVR-L2 é a SEQ ID NO: 88; HVR-L3 é a SEQ ID NO: 89);Clone 184.6.1.N54G (HVR-H1 é a SEQ ID NO: 90; HVR-H2 é a SEQ ID NO: 91; HVR-H3 é a SEQ ID NO: 92; HVR-L1 é a SEQ ID NO: 93; HVR- L2 é a SEQ ID NO: 94; HVR-L3 é a SEQ ID NO: 95);Clone 184.6.1.N54A (HVR-H1 é a SEQ ID NO: 96; HVR-H2 é a SEQ ID NO: 97; HVR-H3 é a SEQ ID NO: 98; HVR-L1 é a SEQ ID NO: 99; HVR- L2 é a SEQ ID NO: 100; HVR-L3 é a SEQ ID NO: 101);Clone 184.6.1.N54Q (HVR-H1 é a SEQ ID NO: 102; HVR-H2 é a SEQ ID NO: 103; HVR-H3 é a SEQ ID NO: 104; HVR-L1 é a SEQ ID NO: 105; HVR-L2 é a SEQ ID NO: 106; HVR-L3 é a SEQ ID NO: 107);Clone 184.6.58.N54S (HVR-H1 é a SEQ ID NO: 108; HVR-H2 é a SEQ ID NO: 109; HVR-H3 é a SEQ ID NO: 110; HVR-L1 é a SEQ ID NO: 111; HVR-L2 é a SEQ ID NO: 112; HVR-L3 é a SEQ ID NO: 113);Clone 184.6.58.N54G (HVR-H1 é a SEQ ID NO: 114; HVR-H2 é a SEQ ID NO: 115; HVR-H3 é a SEQ ID NO: 116; HVR-L1 é a SEQ ID NO: 117; HVR-L2 é a SEQ ID NO: 118; HVR-L3 é a SEQ ID NO: 119);Clone 184.6.58.N54A (HVR-H1 é a SEQ ID NO: 120; HVR-H2 é a SEQ ID NO: 121; HVR-H3 é a SEQ ID NO: 122; HVR-L1 é a SEQ ID NO: 123; HVR-L2 é a SEQ ID NO: 124; HVR-L3 é a SEQ ID NO: 125);Clone 184.6.58.N54Q (HVR-H1 é a SEQ ID NO: 126; HVR-H2 é a SEQ ID NO: 127; HVR-H3 é a SEQ ID NO: 128; HVR-L1 é a SEQ ID NO: 129; HVR-L2 é a SEQ ID NO: 130; HVR-L3 é a SEQ ID NO: 131);Clone 184.6.1.NS D30E (HVR-H1 é a SEQ ID NO: 143; HVR-H2 é a SEQ ID NO: 144; HVR-H3 é a SEQ ID NO: 145; HVR-L1 é a SEQ ID NO: 140; HVR-L2 é a SEQ ID NO: 141; HVR-L3 é a SEQ ID NO: 142).
[145] As posições dos aminoácidos são numeradas de acordo com o sistema de numeração de Kabat como descrito abaixo.
[146] Figuras 2A e 2B: Ilustram (A) as sequências de aminoácidos das regiões variáveis de cadeia pesada e regiões variáveis de cadeia leve dos anticorpos anti-FGFR3 184.6.1.N54S, 184.6.58, e 184.6.62, e (B) as regiões hipervariáveis de anticorpos anti-FGFR3 1G6, 6G1 e 15B2.
[147] Figuras 3A, 3B e 4: representam de maneira exemplar as sequências da região estrutural de consenso humano aceptora para uso na prática da presente invenção com as sequências identificadas da seguinte forma:
[148] Regiões estruturais de consenso variável pesada (VH) (Fig. 3A, 3B): - região estrutural de consenso VH humano subgrupo Imenos as CDRs de Kabat (SEQ ID NOs: 19, 203-205);- região estrutural de consenso VH humano subgrupo Imenos regiões hipervariáveis alongadas (SEQ ID NOS:20 e 206-208, 21 e 209211, 22 e 212-214);- região estrutural de consenso VH humano subgrupo IImenos as CDRs de Kabat (SEQ ID NOs: 23, 215-217);- região estrutural de consenso VH humano subgrupo IImenos regiões hipervariáveis alongadas (SEQ ID NOS:24 e 218-220, 25 e 221223, 26 e 224-226);- região estrutural de consenso VH humano subgrupo II menos regiões alongadas;- região estrutural de consenso VH humano subgrupo III menos as CDRs de Kabat (SEQ ID NOs: 27, 227-229);- região estrutural de consenso VH humano subgrupo III menos regiões hipervariáveis alongadas (SEQ ID NOS:28 e 230-232, 29 e 233235, 30 e 236-238);- região estrutural VH humana aceptora menos as CDRs de Kabat (SEQ ID NOs: 31, 239-241);- região estrutural VH humana aceptora menos regiões hipervariáveis alongadas (SEQ ID NOS: 32 e 242-244, 33 e 2245-247);- região estrutural VH humana aceptora 2 menos as CDRs de Kabat (SEQ ID NOs: 34, 248-250);- região estrutural VH humana aceptora 2 menos regiões hipervariáveis alongadas (SEQ ID NOS:35 e 251-253, 36 e 254-256, 37 e 257259).
[149] Regiões estruturais de consenso variável leve (vl) (fig. 4):- região estrutural de consenso VL kappa humano subgrupo I (Kv1): (SEQ ID NO: 38 e 260-262);- região estrutural de consenso VL kappa humano subgrupo II (SEQ ID NO: 39 e 263-265);- região estrutural de consenso VL kappa humano subgrupo III (SEQ ID NO: 40 e 266-268);- região estrutural de consenso VL kappa humano subgrupo IV (SEQ ID NO: 41 e 269-271).
[150] Figura 5: descreve as sequências da região estrutural (ou do arcabouço (framework)) da cadeia leve (SEQ ID NOS: 42-45) e cadeia pesada (SEQ ID NOS: 46, 47, 175, 176) do huMAb4D5-8. Os números em sobrescrito/negrito indicam as posições de aminoácidos de acordo com Kabat.
[151] Figura 6: descreve as sequências da região estrutural (ou do arcabouço (framework)) da cadeia leve (SEQ ID NOS: 42, 43, 177, 45) e cadeia pesada (SEQ ID NOS: 46, 47, 178 e 176) do huMAb4D5-8. Os números em sobrescrito/negrito indicam as posições de aminoácidos de acordo com Kabat.
[152] Figura 7: O silenciamento gênico (knockdown) de FGFR3 em células RT112 de câncer de bexiga inibe a proliferação e induz a parada do ciclo celular em G1 em ensaios in vitro, e suprime o crescimento tumoral em ensaios in vivo. Três diferentes RNAs curtos (shRNAs) para FGFR3 foram clonados em um vetor de expressão induzível por Tet. As células RT112 expressando estavelmente os shRNAs de FGFR3 ou um shRNA controle foram estabelecidas usando a seleção com puromicina. (A) blots representativos exibindo a expressão de FGFR3 em clones selecionados tratados com ou sem doxiciclina (Dox, 0, 0,1 e 1 μg/ml, da esquerda para a direita). (B) incorporação de [H3]-timidina pelas células RT112 estáveis. Os clones de células RT112 estáveis foram cultivados com ou sem 1 μg/ml de doxiciclina durante 3 dias antes das 16 horas de incubação com [H3]-timidina (1 μCi por poço). As contagens de [H3]-timidina incorporadas foram normalizadas a partir de células sem a indução com doxiciclina. As barras de erro representam EPM. (C) Histogramas obtidos pela citometria de fluxo por fluorescência de células RT112 estáveis. Os clones RT112 expressando shRNA controle ou shRNA4 FGFR3 foram cultivados com ou sem 1 μg/ml de doxiciclina por 72 horas, e os núcleos foram corados com iodeto de propídio (PI). Resultados semelhantes foram obtidos para shRNA2 e 6 de FGFR3 (Figura 16). (D) O crescimento de células RT112 que expressam shRNA controle (n = 9 por grupo de tratamento) ou shRNA4 FGFR3 (n = 11 por grupo de tratamento) em camundongos. Os camundongos receberam 5% de sacarose sozinha ou suplementada com 1 mg/ml de doxiciclina, e o tamanho do tumor foi mensurado duas vezes por semana. As barras de erro representam EPM. Resultados semelhantes foram obtidos para o shRNA2 e 6 FGFR3 (Figura 16). Painel Inferior: Expressão de proteína FGFR3 em lisados de células de tumores extraídas a partir de xenoenxertos com células shRNA controle ou shRNA4 FGFR3 estáveis.
[153] Figura 8: O R3Mab bloqueia a interação FGF/FGFR3. (A) ligação seletiva do FGFR3 humano pelo R3Mab. Proteínas quiméricas FGFR1- 4 Fc humanas foram imobilizadas e incubadas com uma quantidade crescente de R3Mab. A ligação específica foi detectada utilizando um anticorpo anti-Fab humano. (BC) Bloqueio da ligação do FGF1 ao FGFR3-IIIb (B) ou IIIc (C) humano pelo R3Mab. A ligação específica foi detectada usando um anticorpo policlonal específico para FGF1 biotinilado. (D-E) Bloqueio da ligação do FGF9 ao FGFR3- IIIb (D) ou IIIc (E) humano pelo R3Mab. A ligação específica foi detectada usando um anticorpo policlonal específico para FGF9 biotinilado. As barras de erro representam o erro padrão da médio (EPM) e são, por vezes, menores do que os símbolos.
[154] Figura 9: O R3Mab inibe a proliferação celular Ba/F3 induzida pelo FGFR3 tipo selvagem e mutante. (A) O efeito inibitório de R3Mab sobre a viabilidade de células Ba/F3 que expressam FGFR3-IIIb humano tipo selvagem. As células foram cultivadas em meio sem FGF1 (sem FGF1), ou na presença de 10 ng/ml de FGF1 mais 10 μg/ml de heparina isoladamente (FGF1), ou em combinação com um anticorpo de controle (Controle) ou R3Mab. A viabilidade celular foi avaliada com o CellTiter-Glo (Promega) após 72 hrs de incubação com os anticorpos. (B) inibição de FGFR3 e da fosforilação da MAPK pelo R3Mab em células estáveis Ba/F3-FGFR3-IIIbWT. As células foram tratadas com 15 ng/ml de FGF1 e 10 μg/ml de heparina (+) ou apenas heparina (-) por 10 minutos, após a pré-incubação com Ac (anticorpo) controle(Ctrl), diminuindo a quantidade de R3Mab (1, 0,2, 0,04 μg/ml, respectivamente) em PBS, ou PBS sozinho (Mock) por 3 horas. Os lisados foram submetidos ao ensaio de imunoblotting para avaliar a fosforilação do FGFR3 e p44/42 MAPK com anticorpos para pFGFRY653/654 e pMAPKTre202/Tir204, respectivamente. (C) Representação esquemática das localizações preferenciais (hot spots) e frequência de mutação do FGFR3 no câncer de bexiga (numeração da sequência descrita é baseada na sequência de aminoácidos da isoforma FGFR3-IIIb), com base em dados publicados (32). TM, domínio transmembrana; TK1 e TK2, domínio da tirosina quinase 1 e 2. (D-H) O efeito inibitório de R3Mab sobre a viabilidade de células Ba/F3 expressando FGFR3 mutante associado ao câncer. O G372C é derivado da isoforma IIIc, e os restantes são derivados da isoforma IIIb. A numeração das sequencias para todos os mutantes se baseia na sequência de aminoácidos da isoforma FGFR3 IIIb (incluindo o mutante G372C, que seria numerado como G370C se fosse baseado na sequência de aminoácidos da isoforma FGFR3 IIIc). A viabilidade celular foi avaliada após 72 horas de incubação com os anticorpos, conforme descrito em (A). As barras de erro representam EPM.
[155] Figura 10: Mapeamento de epítopos para R3Mab e a estrutura cristalina do complexo entre o fragmento Fab R3Mab e IgD2-D3 do FGFR3-IIIb humano. (A) Epítopo determinado pela ligação de 13 peptídeos abrangendo a IgD2-D3 do FGFR3 humano ao R3Mab. Cada peptídeo biotinilado foi capturado em poços de placas de microtitulação revestidos com estreptavidina e incubado com R3Mab. A ligação específica de R3Mab foi detectada usando um anticorpo de cabra anti-IgG humano. (B) Alinhamento da sequência de peptídeos do FGFR3 humano 3 (LAVPAANTVRFRCPA (SEQ ID NO: 179)) e 11 (SDVEFHCKVYSDAQP (SEQ ID NO: 180) com segmentos extracelulares do FGFR1 humano peptídeo 3: HAVPAAKTVKFKCPS (SEQ ID NO:181); peptídeo 11: SNVEFMCKVYSDPQP (SEQ ID NO:182)). Os resíduos FGFR1 envolvidos na interação FGF2-FGFR1, ligação de heparina, e associação receptor-receptor são exibidos com fontes em negrito, em itálico e sublinhadas, respectivamente. Atribuição funcional dos resíduos FGFR1 é baseada em Plotnikov et al. (34). (C) Estrutura do R3Mab Fab (mostrado na fita- hélice, cadeia leve em cinza, cadeia pesada em preto) no complexo com FGFR3 humano IgD2-D3 (mostrado na superfície molecular, em branco). Os resíduos de receptor envolvido na ligação do ligante e dimerização são coloridos em cinza hachurado e cinza escuro, respectivamente com base em Plotnikov et al. (34). (D) Uma visão aproximada da estrutura cristalina mostra que a CDR-H3 e H2 do Fab constituem os principais sítios de interação com IgD2 e IgD3 do FGFR3. (E) Superposição do complexo FGFR3-IIIc-FGF1 (PDB código 1RY7) com o complexo FGFR3-IIIb-Fab. O FGFR3-IIIc e FGF1 são coloridos em cinza e cinza escuro, respectivamente. O FGFR3-IIIb é exibido em branco e o Fab é exibido em cinza claro para a cadeia leve, e cinza escuro para a cadeia pesada. Foi usado a IgD2 como âncora para a superposição. Observe o IgD2 bem sobreposta a partir de ambas as estruturas e a nova conformação adotada pela IgD3 do FGFR3-IIIb quando ligado pelo R3Mab. (F) Outra representação da superposição do complexo FGFR3-IIIc-FGF1 (PDB código 1RY7) com o complexo FGFR3-IIIb-Fab. FGFR3-IIIc e FGF1 são mostrados como superfícies moleculares que estão em textura quadriculada/cinza e textura pontilhada/cinza escuro, respectivamente. O FGFR3-IIIb é exibido em branco e o Fab é exibido em cinza para a cadeia leve, e preto para a cadeia pesada. Foi usado a IgD2 como âncora para a superposição. Observe o IgD2 bem sobreposta a partir de ambas as estruturas e a nova conformação adotada pela IgD3 do FGFR3-IIIb quando ligado pelo R3Mab.
[156] Figura 11: R3Mab inibe a proliferação, crescimento clonal e sinalização de FGFR3 em células de câncer da bexiga expressando FGFR3S249C mutante ou tipo selvagem. (A) Inibição da incorporação de [H3]- timidina por R3Mab na linhagem de célula de câncer de bexiga RT112. As barras de erro representam EPM. (B) Bloqueio da sinalização FGFR3 ativada por FGF1 pelo R3Mab (15 μg/ml) na linhagem de célula de câncer de bexiga RT112 comparado com o meio de tratamento sozinho (Mock) ou anticorpo controle (Ctrl). Os lisados celulares foram imunoprecipitados com o anticorpo anti-FGFR3 e avaliados quanto à fosforilação de FGFR3 com um anticorpo anti-fosfotirosina (4G10). O lisados foram submetidos à técnica de immunoblotting para detectar a fosforilação da AKT (pAKTS473) e MAPK p44/42 (pMAPKTre202/Tir204). (C) Inibição do crescimento clonal pelo R3Mab (10 μg/ml) na linhagem de célula de câncer de bexiga UMUC-14 (carregando FGFR3S249C) em comparação com o meio de tratamento sozinho (Mock) ou anticorpo controle (Ctrl). (D) A determinação quantitativa do estudo em (C) informando o número de colônias maiores que 120 μm de diâmetro por poço a partir de uma replicata de 12 poços. As barras de erro representam EPM. P < 3,4 X 10-9 contra os grupos Mock ou Ctrl. (E) Inibição da fosforilação de FGFR3 em células UMUC-14 pelo R3Mab (15 μg/ml). A fosforilação do FGFR3 foi analisada como em (B). Deve-se observar a fosforilação constitutiva do FGFR3 nesta linhagem celular.
[157] Figura 12: R3Mab diminui o nível de estado estacionário do dímero FGFR3S249C ligado à dissulfeto conduzindo o equilíbrio dímero- monômero em direção ao estado monomérico. (A) Efeito do R3Mab sobre o dímero FGFR3S249C em células UMUC-14. As células foram incubadas com R3Mab (15 μg/ml) ou um anticorpo controle (Ctrl) por 3 horas, e lisados de células inteiras foram analisados por immunoblotting em condições não redutoras e condições redutoras. (B) Efeito do bloqueador DTNB sem sulfidrila sobre o equilíbrio dímero-monômero FGFR3S249C em células UMUC-14. As células UMUC -14 foram tratadas com concentrações crescentes de DTNB por 3 horas, e os lisados celulares foram analisados como em (A). (C) Efeito do R3Mab sobre o dímero FGFR3S249C recombinante purificado em ensaio in vitro. O dímero FGFR3S249C composto por IgD2-D3 foi purificado através de uma coluna por exclusão de tamanho, e incubado com PBS (Mock), anticorpo controle (Ctrl), ou R3Mab a 37°C. As amostras foram coletadas no momento indicado para análise por immunoblotting em condições de não redução. O dímero-monômero FGFR3 foi detectado utilizando o anticorpo anti-FGFR3 do hibridoma 6G1 (A-C).
[158] Figura 13: O R3Mab inibe o crescimento de células de xenoenxerto de câncer de bexiga e o crescimento do enxerto Ba/F3- FGFR3S249C. (A) Efeito do R3Mab sobre o crescimento do xenoenxerto de células de câncer de bexiga RT112 pré-estabelecidas em comparação com o tratamento com controle (apenas veículo). n = 10 por grupo. (B) Inibição da sinalização de FGFR3 em tecidos tumorais RT112 pelo R3Mab. Em uma experiência separada, os tumores RT112 xenoenxertados que foram tratados com 15 μg/kg de anticorpo controle (Ctrl) ou R3Mab por 48 horas ou 72 horas foram coletados (n = 3 por grupo), homogeneizados e analisados quanto à ativação de FRS2a e MAPK por immunoblot. (C) Efeito do R3Mab sobre o crescimento da aloenxertos Ba/F3-FGFR3S249C pré-estabelecidos. n = 10 por grupo. (D) Efeito do R3Mab sobre o crescimento da xenoenxertos UMUC-14 de câncer de bexiga pré-estabelecidos, n = 10 por grupo. (E) Efeito do R3Mab sobre o dímero FGFR3S249C e sinalização em tecidos de tumor UMUC -14. Tumores de xenoenxertos UMUC-14 que foram tratados com 30 mg/kg de anticorpo controle (Ctrl) ou R3Mab por 24 horas ou 72 horas foram coletados (n = 3 por grupo), homogeneizados e analisados quanto ao dímero-monômero FGFR3S249C, bem como a ativação da MAPK por immunoblot. O dímero- monômero FGFR3 foi detectado utilizando um anticorpo policlonal de coelho anti-FGFR3 sc9007 para evitar a interferência com as IgGs de camundongos a partir dos lisados de tumor. As barras de erro representam EPM.
[159] Figura 14: A ADCC contribui para a eficácia antitumoral do R3Mab em modelos de mieloma múltiplo positivos para t(4; 14). (A-B) Efeito do R3Mab sobre o crescimento de xenoenxertos de mieloma pré-estabelecidos OPM2 (A) e KMS11 (B). n = 10 por grupo. (C-F) citólise da linhagem de células de mieloma OPM2 (C) e KMS11 (D), ou da linhagem de células de câncer de bexiga RT112 (E) e UMUC -14 (F) induzida pelo R3Mab em cultura de células. As células de câncer mieloma ou de bexiga foram incubadas com células mononucleares do sangue periférico (PBMC) humanas recém isoladas na presença de um anticorpo R3Mab ou controle. A citotoxicidade foi determinada mensurando o LDH liberado no sobrenadante. (G-H) Efeito do R3Mab ou seu DANA mutante sobre o crescimento da xenoenxertos de mieloma pré- estabelecidos OPM2 (G) e KMS11 (H); n = 10 por grupo. Barras de erro representam EPM e por vezes são menores do que os símbolos.
[160] Figura 15: O silenciamento gênico (knockdown) do FGFR3 utilizando siRNA inibe a proliferação celular de linhagens de células de câncer de bexiga. Seis a sete siRNAs para FGFR3 diferentes e três siRNAs não específicos usados como controle foram projetados e sintetizados na Genentech. As linhagens de células de câncer de bexiga RT112 (A), SW780 (B), RT4 (C) e UMUC-14 (D) foram semeadas em placas de 96 poços (3000 células por poço) e deixadas em repouso para aderirem às placas durante a noite, e em seguida foram transfectadas transitoriamente com 25 nM de siRNA no complexo com RNAiMax (Invitrogen). 72 horas após a transfecção, foi adicionado à cultura (A, C e D) [H3]-timidina (1 μCi por poço) e a incubação seguiram por mais 16 horas. A [H3]-timidina incorporada foi quantificada com TopCount. Os dados foram normalizados a partir das células transfectadas apenas com RNAiMax (Mock). As barras de erro representam EPM. Painel inferior: blots representativos mostrando a expressão de FGFR3 em células transfectadas com siRNA. (B) A viabilidade celular foi mensurada utilizando o CellTiter-Glo (Promega) 96 horas após a transfecção. As barras de erro representam EPM.
[161] Figura 16: O silenciamento gênico (knockdown) de FGFR3 na linhagem de células de câncer de bexiga RT112 induz a parada do ciclo celular em G1 em ensaio in vitro, e suprime o crescimento do tumor em ensaio in vivo. Três diferentes RNAs para FGFR3 foram desenvolvidos e clonados em um vetor retroviral para a expressão de shRNA induzível por Tet. Os clones RT112 expressando estavelmente os shRNAs de FGFR3 ou shRNA controle foi estabelecido usando a seleção com puromicina. (A) histogramas da citometria de fluxo fluorescente do DNA de núcleos corados com iodeto de propídio (PI) obtidos a partir de células RT112 expressando de maneira estável shRNA2 ou shRNA6 para FGFR3 após o tratamento com ou sem 1 μg/ml de doxiciclina por 72 horas. (B) O crescimento de células RT112 expressando de maneira estável shRNA2-4 para FGFR3 (n = 11 por grupo de tratamento) ou shRNA6-6 FGFR3 (n = 10 por grupo de tratamento) em camundongos nu/nu. Os camundongos portadores de tumor receberam apenas 5% de sacarose (círculo) ou 5 % de sacarose, acrescido de 1 mg/ml de doxiciclina (quadrado preenchido), e os tumores foram mensurados com paquímetro duas vezes por semana. As barras de erro representam EPM.
[162] Figura 17: Efeito de anticorpos anti-FGFR3 hibridoma 16G, 6G1 e 15B2 sobre a proliferação celular de Ba/F3 induzida pelo FGFR3 do tipo selvagem e mutante. Anticorpos anti-FGFR3 de hibridoma foram gerados pela imunização de camundongos BALB/c com FGFR3-IIIb/Fc humano ou FGFR3- IIIc humano/Fc quimérico. As células de hibridoma fusionado foram selecionadas utilizando a seleção por hipoxantina-aminopterina-timidina em meio D a partir do kit de seleção de hibridoma ClonaCell® (StemCell Technologies, Inc., Vancouver, BC, Canadá). Os anticorpos de hibridoma foram sequencialmente selecionados por ELISA pelas suas capacidades de se ligarem ao FGFR3-IIIb e FGFR3-IIIc e por FACS pela capacidade de reconhecerem o FGFR3 na superfície celular. Os hibridomas selecionados foram então clonados por diluição limitante. 16G, 6G1 e 15B2 são clones utilizados para avaliar o efeito sobre a proliferação de células Ba/F3 que expressam FGFR3 tipo selvagem ou mutado da mesma forma conforme descrito na Figura 9A. As barras de erro representam EPM.
[163] Figura 18: Comparação de epítopos R3Mab determinado pelo mapeamento de peptídeos e análise da estrutura cristalina. (A) Epítopo revelado pela estrutura do fragmento Fab do R3Mab no complexo com o segmento IgD2-D3 extracelular do FGFR3 humano. Os resíduos FGFR3 contatados com a cadeia pesada e cadeia leve do Fab estão coloridos em preto e cinza, respectivamente. (B) Local dos peptídeos 3 e 11 no FGFR3.
[164] Figura 19: R3Mab inibe a proliferação e sinalização de FGFR3 em células de câncer de bexiga contendo FGFR3S249C tipo selvagem ou mutante. (A) Inibição da viabilidade celular pelo R3Mab na linhagem de células de câncer de bexiga RT4. A viabilidade celular foi avaliada com o CellTiter-Glo (Promega) após 96 hrs de incubação com o anticorpo. As barras de erro representam EPM. (B) Bloqueio pelo R3Mab (15 μg/ml) da sinalização de FGFR3 ativada por FGF1 na linhagem de células de câncer de bexiga RT4. (C) Inibição da incorporação de [H3]-timidina pelo R3Mab na linhagem de célula de câncer de bexiga RCC-97-7 (contendo FGFR3S249C). As barras de erro representam EPM. (D) Inibição da fosforilação de FGFR3 em células TCC-97-7 pelo R3Mab (15 μg/ml). (E) Diminuição do dímero FGFR3S249C em células TCC- 97-7 após 3 horas de incubação com R3Mab (15 μg/ml) em comparação com um anticorpo controle (Ctrl).
[165] Figura 20: Efeito de inibidores da endocitose sobre a internalização de R3Mab e dímero FGFR3S249C em células UMUC-14. (A) Efeito de inibidores da endocitose sobre a internalização de R3Mab. As células UMUC -14, pré-tratadas com vários inibidores de endocitose ou DMSO por 1 hora a 37 °C foram incubadas com R3Mab (15 μg/ml) durante 3 horas a 37 °C para permitir a interiorização. Uma lavagem em pH baixo foi usada para remover o R3Mab da superfície celular para visualizar os anticorpos internalizado. As células foram fixadas e coradas com anti-IgG humano marcado com Alexa 488. A imagem foi obtida por meio de microscopia confocal. (D) Efeito de inibidores da endocitose sobre o dímero FGFR3S249C em células UMUC-14 tratadas com R3Mab. As células UMUC -14, pré-tratadas com vários inibidores de endocitose ou DMSO por 1 hora a 37 °C foram incubadas com Mock (pista 1), anticorpo controle (Pista 2), ou R3Mab (15 μg/ml, pista 3) por 3 horas a 37 °C. Os lisados celulares foram analisados quanto às proteínas FGFR3 sob condições não redutoras ou condições redutoras pela técnica de immunoblotting. Note-se que a clorpromazina (inibidor da endocitose mediada por clatrina) e genisteína (pan- inibidor da endocitose) bloqueou a internalização de R3Mab, mas não teve efeito sobre a diminuição do dímero FGFR3S249C induzida por R3Mab.
[166] Figura 21: Sensibilidade de detecção de diferentes anticorpos anti-FGFR3 em relação a FGFR3S249C monomérico e dimérico sob condições não redutoras. As células UMUC-14 foram lisadas após o tratamento com R3Mab (pista 1), IgG1 controle (pista 2) ou PBS (Pista 3) por 3 horas, e lisados os celulares foram submetidos a análises por immunoblotting sob condições redutoras e não redutoras. Deve-se observar que o 6G1 (anticorpo de hibridoma murino gerado na Genentech) detectou FGFR3S249C na forma de dímero e monômero, enquanto o sc9007 (anticorpo policlonal de coelho, Santa Cruz Biotechnology) ou sc13121 (anticorpo de hibridoma murino, Santa Cruz Biotechnology), detectou preferencialmente o FGFR3S249C dimérico.
[167] Figura 22: Efeito do R3Mab sobre a proliferação de células de mieloma múltiplo t(4; 14)+. (A) Efeito inibitório do R3Mab sobre a incorporação de [H3]-timidina pelas células UTMC-2. As células UTMC-2 foram cultivadas em meio contendo R3Mab ou anticorpo controle, na presença de 25 ng/ml de FGF9 e 5 μg/ml de heparina ou apenas heparina (sem FGF9). Após 6 dias de incubação, foi adicionado [H3]-timidina por 16 hr de incubação. Os dados foram normalizados com as células cultivadas na ausência de FGF9 e anticorpos. (BC) Efeito do R3Mab sobre a incorporação de [H3]-timidina pelas células OPM2 (B) e KMS11 (C). As células cultivadas em meio contendo 1,5% de FBS foram tratadas com R3Mab ou anticorpo controle durante 6 dias. Os dados foram normalizados a partir de células não tratadas. As barras de erro representam EPM.
[168] Figura 23: Níveis de expressão de FGFR3 na superfície celular em células de mieloma e de câncer de bexiga. (A) A expressão de FGFR3 na superfície celular em células de mieloma e células de câncer de bexiga foi avaliada pela análise de FACS. As células foram marcadas com anticorpo monoclonal (Mab) de camundongo conjugado com ficoeritina contra FGFR3 humano (FAB766P, R&D Systems) ou com controle isotópico IgG1 de camundongo conjugado com ficoeritina (BD PharMingen). (B) Análise de Scatchard da densidade de FGFR3 em células de mieloma e células de câncer de bexiga. O R3Mab foi radioiodinado, e incubado com as células em suspensão com anticorpo não marcado em excesso. Após a incubação em temperatura ambiente por 2 horas, as células foram peletizadas por centrifugação e lavadas duas vezes. O I125 especificamente ligado foi determinado. Densidade do receptor e a afinidade de ligação (Kd) representam a média de dois experimentos de ligação.
[169] Figura 24: Efeito do R3Mab ou seu mutante DANA no crescimento do xenoenxerto de células de carcinoma de bexiga. (A) Efeito do R3Mab e seus mutantes DNA (50 mg/kg cada) sobre o crescimento de tumores RT112 pré-estabelecidos. (B) Efeito do R3Mab e seus mutantes DANA (50 mg/kg cada) sobre o crescimento de tumores UMUC-14 pré-estabelecidos. As barras de erro representam EPM.DESCRIÇÃO DETALHADA DA INVENÇÃO
[170] A presente invenção fornece anticorpos anti-FGFR3 que são úteis para, por exemplo, o tratamento ou prevenção de estados de doenças associados com a expressão e/ou atividade de FGFR3, tal como o aumento da expressão e/ou atividade ou expressão indesejada e/ou atividade. Em algumas realizações, os anticorpos da presente invenção são usados para tratar um tumor, um câncer, e/ou distúrbio da proliferação celular.
[171] Em outro aspecto, os anticorpos anti-FGFR3 da presente invenção encontram utilidade como reagentes para detecção e/ou isolamento de FGFR3, tal como a detecção de FGFR3 em vários tipos de tecidos e células.
[172] A presente invenção fornece ainda métodos de fazer e utilizar anticorpos anti-FGFR3, e polinucleotídeos que codificam os anticorpos anti-FGFR3.TÉCNICAS GERAIS
[173] As técnicas e procedimentos descritos ou mencionados no presente são de modo geral bem compreendidos e comumente empregados utilizando a metodologia convencional pelos técnicos hábeis no assunto, como, por exemplo, as metodologias amplamente utilizadas e descritas em Sambrook et al., Molecular Cloning: A Laboratory Manual 3°. Edição (2001) Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY. CURRENT PROTOCOLS IN MOLECULAR BIOLOGY (F. M. Ausubel, et al. (2003)); da série METHODS IN ENZYMOLOGY (Academic Press, Inc.): “PCR 2: A PRACTICAL APPROACH” (M. J. MacPherson, B. D. Hames e G. R. Taylor. (1995)), Harlow & Lane, eds. (1988) “ANTIBODIES, A LABORATORY MANUAL, and ANIMAL CELLCULTURE” (R. I. Freshney. (1987)).DEFINIÇÕES
[174] Um anticorpo “isolado” é aquele que foi identificado, separado e/ou recuperado a partir de um componente do seu ambiente natural. Os componentes contaminantes do seu ambiente natural são materiais que poderiam interferir no uso diagnóstico ou terapêutico para o anticorpo e podem incluir enzimas, hormônios e outros solutos proteináceos ou não-proteináceos. Em exemplos de realizações preferidas, o anticorpo será purificado (1) até mais de 95% em peso de anticorpo, conforme determinado através do método Lowry e, de maior preferência, mais de 99% em peso; (2) até grau suficiente para a obtenção de pelo menos 15 resíduos de sequência de aminoácidos interna ou N-terminal, por meio do uso de um sequenciador do tipo “spinning cup”; ou (3) até a homogeneidade através de SDS-PAGE sob condições redutoras ou não redutoras utilizando azul de Coomassie™ ou, preferencialmente, coloração de prata. O anticorpo isolado inclui o anticorpo in situ em células recombinantes, desde que pelo menos um componente do ambiente natural do anticorpo não esteja presente. Normalmente, entretanto, o anticorpo isolado é preparado através de pelo menos uma etapa de purificação.
[175] Uma molécula "isolada" de ácido nucleico é uma molécula de ácido nucleico que é identificada e separada de pelo menos uma molécula de ácido nucleico contaminante com a qual está comumente associada à fonte natural de ácido nucleico. Uma molécula de ácido nucleico isolada é diferente, sob a forma ou a configuração em que é encontrado na natureza. Moléculas de ácido nucleico isoladas são moléculas, por conseguinte, distintas das moléculas de ácidos nucleicos naturais, uma vez que elas existem em células. No entanto, uma molécula de ácido nucleico isolada inclui uma molécula de ácido nucleico contida nas células que normalmente expressam o ácido nucleico (por exemplo, ácido nucleico que codifica o anticorpo), no qual, por exemplo, a molécula de ácido nucleico está localizada em um cromossomo diferente da encontrada na célula natural.
[176] A expressão “resíduos do domínio variável como na numeração de Kabat" ou "posição dos aminoácidos como na numeração de Kabat", e variantes dos mesmos, referem-se ao sistema de numeração utilizado para o domínio variável da cadeia pesada ou cadeia leve da compilação dos anticorpos em Kabat et al., Sequences of Proteins of Immunological Interest, 5a Ed. National Institutes of Health, Bethesda, MD (1991). Usando este sistema de numeração, a sequência de aminoácido linear real pode conter menos ou mais aminoácidos, o que corresponde à redução ou inserção em uma FR ou CDR do domínio variável. Por exemplo, um domínio variável de cadeia pesada pode incluir um único aminoácido inserido (resíduo 52A, de acordo com Kabat) após o resíduo 52 da H2 e resíduos inseridos (por exemplo, resíduos 82a, 82b, 82c, etc., de acordo com Kabat), após o resíduo 82 da FR de cadeia pesada. A numeração de Kabat dos resíduos pode ser determinada para um dado anticorpo pelo alinhamento nas regiões de homologia da sequência do anticorpo com uma sequência numerada de Kabat "padrão".
[177] As frases “substancialmente semelhante”,“substancialmente o mesmo", conforme utilizadas no presente, denotam um grau de semelhança suficientemente elevado entre dois valores numéricos (geralmente, uma associada a um anticorpo da presente invenção e a outra associada a um anticorpo de referência/comparação) de tal forma que um técnico hábil no assunto iria considerar a diferença entre os dois valores como de pouca ou nenhuma significância biológica e/ou estatística no contexto da característica biológica mensurada pelos valores citados (por exemplo, valores Kd). A diferença entre os dois valores é, por exemplo, inferior a 50%, preferencialmente menor que 40%, preferencialmente inferior a 30%, preferencialmente menor que 20% e/ou preferencialmente inferior a 10% em função do valor do anticorpo de referência/comparação.
[178] “Afinidade de ligação” refere-se, em geral, à força da soma total de interações não covalentes entre um único sítio de ligação de uma molécula (tal como anticorpo) e seu parceiro de ligação (tal como, um antígeno). A “afinidade de ligação” a menos que indicado de outro modo, refere-se à afinidade de ligação intrínseca que reflete uma interação de 1:1 entre os membros do par ligante (por exemplo, anticorpo e antígeno). A afinidade da molécula X para seu parceiro Y pode geralmente ser representada pela constante de dissociação (Kd). Desejavelmente a Kd é de 1 x 10-7, 1 x 10-8, 5 x 10-8, 1 x 10-9, 3 x 10-9, 5 x 10-9, ou mesmo 1 x 10-10 ou mais forte. A afinidade pode ser medida através de métodos conhecidos no estado da técnica, incluindo os métodos descritos na presente invenção. Anticorpos de baixa afinidade ligam de forma fraca o antígeno e tendem a dissociar rapidamente, enquanto os anticorpos de alta afinidade ligam o antígeno de forma mais forte e permanecem ligados por mais tempo. Uma variedade de métodos para mensurar a afinidade de ligação é conhecida no estado da técnica, no qual qualquer um destes métodos pode ser utilizado para os propósitos da presente invenção. Exemplos de realização especificamente ilustrativos são descritos a seguir.
[179] Em um exemplo de realização, o "Kd" ou "valor de Kd" de acordo com a presente invenção é mensurado pela realização de um teste de ligação com antígeno radiomarcado (RIA) com a versão Fab de um anticorpo de interesse e seus antígenos conforme descrito pelo seguinte ensaio que mensura a afinidade de ligação em solução de Fabs para antígenos pelo equilíbrio do Fab com uma concentração mínima de antígeno marcado com I125, na presença de uma titulação seriada de antígeno não marcado e, em seguida, capturando os antígenos ligados com uma placa revestida com anticorpos anti-Fab (Chen, et al., (1999) J. Mol Biol 293:865-881). Para estabelecer as condições para o ensaio, placas de microtitulação (Dynex) são revestidas durante a noite com 5 μg/ml de anticorpo anti-Fab (Cappel Labs), em 50 mM de carbonato de sódio (pH 9,6) e, em seguida, é feito o bloqueio com albumina bovina 2% (p/v) em PBS por duas a cinco horas à temperatura ambiente (cerca de 23 °C). Em uma placa não adsorvente (Nunc # 269620), 100 pM ou 26 pM de antígeno-[I125] são misturados com diluições seriadas de um Fab de interesse (por exemplo, coerente com a avaliação do anticorpo anti-VEGF, Fab-12 em Presta et al., (1997) Cancer Res. 57:4593-4599) (57:4593-4599). A Fab de interesse é, em seguida, incubada durante a noite, no entanto, a incubação pode continuar por um período maior (por exemplo, cerca de 65 horas) para garantir que o equilíbrio seja alcançado. Posteriormente, as misturas são transferidas para a placa de captura para a incubação em temperatura ambiente (por exemplo, por uma hora). A solução é então retirada e a placa lavada oito vezes, com 0,1% de Tween-20 em PBS. Após as placas estarem secas, 150 μl/poço de cintilante (MicroScint-20; Packard) é adicionado, e as placas são contadas em um contador TOPCOUNT gamma (Packard) por dez minutos. As concentrações de todos os Fab que resulta em menor ou igual a 20% do máximo de ligação são escolhidas para a utilização em ensaios de ligação competitiva. De acordo com outro exemplo de realização, Kd ou valor de Kd é mensurado pelo ensaio de ressonância plasmônica de superfície utilizando um BIAcoreTM-2000 ou BIAcoreTM-3000 (BIAcore, Inc., Piscataway, NJ) a 25 °C com chips CM5 de antígeno imobilizado 10 Unidades de Resposta (RU). Resumidamente, chips biossensores de dextrano carboximetilados (CM5, BIAcore Inc.) são ativados com cloridrato de N-etil-N'-(3-dimetilaminopropil)-carbodiimida (EDC) e N- hidroxisucinimida (NHS) de acordo com as instruções do fornecedor. O antígeno é diluído com 10 mM de acetato de sódio, pH 4,8, em 5 μg/ml (~ 0,2 μM) antes de ser injetado a uma velocidade de fluxo de 5 μl/minuto para atingir aproximadamente 10 unidades de resposta (RU) de proteína acoplada. Após a injeção do antígeno, etanolamina 1M é adicionada para bloquear grupos que não reagiram. Para a mediação cinética, diluições seriadas em duplicatas de Fab (0,78 nM e 500 nM) são injetadas em PBS com 0,05% de Tween-20 (PBST) a 25 °C em uma taxa de fluxo de cerca de 25 μl/min. Em alguns exemplos de realização, as seguintes modificações são usadas para o método ensaio da ressonância plasmônica de superfície: o anticorpo é imobilizado ao chip biossensor CM5 para atingir cerca de 400 RU, e para as medições cinéticas, duas diluições seriadas da proteína-alvo (por exemplo, FGFR3-IIIb ou -IIIc) (iniciando a 67 nM) são injetadas em tampão PBST a 25 °C com uma vazão de cerca de 30 μl/minuto. A velocidade de associação (kon) e a velocidade de dissociação (koff) são calculadas utilizando um modelo de ligação simples um- para-um de Langmuir (BIAcore Evaluation Software versão 3.2) pelo ajuste simultâneo de sensogramas de associação e dissociação. A constante do equilíbrio de dissociação (Kd) é calculada como a relação koff/kon. Vide, por exemplo., Chen, Y., et al., (1999) J. Mol. Biol. 293: 865-881. Se a medida for superior a 106 M-1 S-1 pelo ensaio de ressonância plasmônica descrito acima, então a constante de associação pode ser determinada por uma técnica de inibição (quenching) de fluorescência que mede o aumento ou a diminuição das emissões de intensidade de fluorescência (excitação= 295 nm; Emissão = 340 nm, banda de passagem=16 nm) a 25 °C de 20 nM de um anticorpo anti-antígeno (na forma de Fab) em PBS, pH 7,2, na presença de concentrações crescentes do antígeno mensurado em espectrômetro, tal como um espectrofotômetro equipado com válvula do tipo stop-flow (Aviv Instruments) ou um espectrofotômetro SLM-Aminco série 8000 (ThermoSpectronic), com agitador de cuveta.
[180] Uma “constante de associação”, “velocidade de associação" ou "taxa de associação" ou "kon", de acordo a presente invenção também pode ser determinada utilizando o mesmo ensaio de ressonância plasmônica descrito acima usando um BIAcoreTM-2000 ou BIAcoreTM-3000 (BIAcore Inc., Piscataway, NJ) a 25 °C com um chip CM5 de antígeno imobilizado a ~ 10 unidades de resposta (RU). Resumidamente, chips biossensores de dextrano carboximetilados (CM5, BIAcore Inc.) são ativados com cloridrato de N-etil-N'-(3- dimetilaminopropil)-carbodiimida (EDC) e N-hidroxisucinimida (NHS) de acordo com as instruções do fornecedor. O antígeno é diluído com 10 mM de acetato de sódio, pH 4,8, em 5 μg/ml (~ 0,2 μM) antes de ser injetado a uma velocidade de fluxo de 5 μl/minuto para atingir aproximadamente 10 unidades de resposta (RU) de proteína acoplada. Após a injeção do antígeno, etanolamina 1M é adicionada para bloquear grupos que não reagiram. Para a mediação cinética, diluições seriadas em duplicatas de Fab (0,78 nM e 500 nM) são injetadas em PBS com 0,05% de Tween-20 (PBST) a 25 °C em uma taxa de fluxo de cerca de 25 μl/min. Em alguns exemplos de realização, as seguintes modificações são usadas para o método ensaio da ressonância plasmônica de superfície: o anticorpo é imobilizado ao chip biossensor CM5 para atingir cerca de 400 RU, e para as medições cinéticas, duas diluições seriadas da proteína-alvo (por exemplo, FGFR3-IIIb ou -IIIc) (iniciando a 67 nM) são injetadas em tampão PBST a 25 °C com uma vazão de cerca de 30 μl/minuto. A velocidade de associação (kon) e a velocidade de dissociação (koff) são calculadas utilizando um modelo de ligação simples um-para-um de Langmuir (BIAcore Evaluation Software versão 3.2) pelo ajuste simultâneo de sensogramas de associação e dissociação. A constante do equilíbrio de dissociação (Kd) foi calculada como a relação koff/kon. Vide, por exemplo., Chen, Y., et al., (1999) J. Mol. Biol. 293: 865-881. Entretanto, se a constante de associação (on-rate) for superior a 106 M-1 S-1 pelo ensaio de ressonância plasmônica descrito acima, então é preferível que a constante de associação seja determinada por uma técnica de inibição (quenching) de fluorescência que mede o aumento ou a diminuição das emissões de intensidade de fluorescência (excitação= 295 nm; Emissão = 340 nm, banda depassagem=16 nm) a 25 °C de 20 nM de um anticorpo anti-antígeno (na forma de Fab) em PBS, pH 7,2, na presença de concentrações crescentes do antígeno mensurado em espectrômetro, tal como um espectrofotômetro equipado com válvula do tipo stop-flow (Aviv Instruments) ou um espectrofotômetro SLM- Aminco série 8000 (ThermoSpectronic), com agitador de cuveta.
[181] O termo “vetor”, da forma utilizada na presente invenção, refere-se a uma molécula de ácido nucleico capaz de transportar outro ácido nucleico ao qual ele foi ligado. Um tipo de vetor é um “plasmídeo”, que se refere a uma alça (loop) circular de DNA de fita dupla, ao qual, segmentos de DNA adicionais podem ser ligados. Outro tipo de vetor é um vetor fago. Outro tipo de vetor é um vetor viral, em que segmentos de DNA adicionais podem ser ligados dentro do genoma viral. Determinados vetores são capazes de realizar replicação autônoma na célula hospedeira a qual foram introduzidos (por exemplo, vetores bacterianos que possuem uma origem bacteriana de replicação e vetores mamíferos epissomais). Outros vetores (por exemplo, vetores mamíferos não epissomais) podem ser integrados no genoma de uma célula hospedeira mediante a introdução na célula hospedeira, sendo, desta forma, replicado junto com o genoma hospedeiro. Adicionalmente, determinados vetores são capazes de direcionar a expressão de genes aos quais eles estão operacionalmente ligados. Tais vetores são denominados na presente invenção como “vetores de expressão recombinantes” (ou simplesmente, “vetores recombinantes”). Em geral, os vetores de expressão para uso em técnicas de DNA recombinante estão frequentemente presentes na forma de plasmídeos. De acordo com o presente relatório descritivo, os termos “plasmídeo” e “vetor” podem ser utilizados de forma alternada, sendo que o termo “plasmídeo” é a forma de vetor mais comumente utilizada.
[182] “Polinucleotídeo” ou “ácido nucleico”, utilizada na apresente invenção de forma alternada, referem-se a polímeros de nucleotídeos de qualquer comprimento, e incluem DNA e RNA. Os nucleotídeos podem ser desoxirribonucleotídeos, ribonucleotídeos, nucleotídeos modificados ou bases, e/ou análogos destes, ou qualquer substrato que possa ser incorporado em um polímero pela DNA ou RNA polimerase, ou por uma reação sintética. Um polinucleotídeo pode compreender nucleotídeos modificados, tais como nucleotídeos metilados e seus análogos. Se presente, a modificação na estrutura do nucleotídeo pode ser realizada antes ou após a montagem do polímero. A sequência de nucleotídeos pode ser interrompida por componentes não nucleotídeos. Um polinucleotídeo pode ser adicionalmente modificado após a síntese, tal como por conjugação com um marcador. Outros tipos de modificações incluem, por exemplo, "caps", substituição de um ou mais nucleotídeos de ocorrência natural com um análogo, modificações em internucleotídeos tais como, por exemplo, aquelas com ligações não carregadas (por exemplo, metil fosfonatos, fosfotriésteres, fosfoamidatos, carbamatos, etc.) e com ligações carregadas (tal como fosforotioatos, fosforoditioatos, etc.), aquelas que contêm frações pendentes, tais como, por exemplo, proteínas (por exemplo, nucleases, toxinas, anticorpos, peptídeos sinal, ply-L-lisina, etc.), aquelas com intercalantes (tal como acridina, psoraleno, etc.), aquelas que contêm quelantes (tais como, metais, metais radioativos, boro, metais oxidativos, etc.), aquelas que contêm alquilantes, aquelas com ligações modificadas (tal como ácidos nucleicos alfa anoméricos, etc.), bem como as formas não modificadas de polinucleotídeos. Adicionalmente, qualquer um dos grupos hidroxila geralmente presentes nos açúcares podem ser substituídos, por exemplo, por grupos fosfonatos, grupos fosfatos, protegidos por grupos protetores padrão, ou ativados para preparar ligações adicionais a nucleotídeos adicionais, ou podem ser conjugados para suportes sólidos ou semi-sólidos. A extremidade terminal OH 5' e 3' pode ser fosforilada ou substituída com unidades de moléculas orgânicas de proteção ou aminas de 1 a 20 átomos de carbono. Outras hidroxilas podem ainda ser derivatizadas para grupos protetores. Os polinucleotídeos podem ainda conter formas análogas de açúcares ribose ou deoxiribose são geralmente conhecidos na técnica, incluindo, por exemplo, 2'-O- metil-, 2'-O-alil, 2'-fluoro- ou 2'-azido-ribose, análogos de açúcar carboxílico, açúcares a-anoméricos, açúcares epiméricos tais como arabinose, xiloses ou lixoses, açúcares piranose, açúcares furanose, sedoheptuloses, análogos acíclicos, e análogos de nucleosídeos abásicos tais como metil-ribosídeo. Uma ou mais das ligações fosfodiéster podem ser substituídas por grupos de ligação alternativos. Estes grupos de ligação alternativos incluem, mas não se limitam a, realizações em que o fosfato é substituído por P(O)S("tioato"), P(S)S ("ditioato"), "(O)NR2” ("amidato"), P(O)R, P(O)OR', CO ou CH2 ("formacetal"), em que cada R ou R' é independentemente H ou alquila substituída ou não-substituída (1-20 C.) contendo opcionalmente uma ligação éter (-O-), arila, alquenila, cicloalquila, cicloalquenila ou araldila. Nem todas as ligações no polinucleotídeo precisam ser idênticas. A descrição anterior aplica-se a todos os polinucleotídeos descritos no presente, incluindo RNA e DNA.
[183] "Oligonucleotídeo," da forma utilizada no presente, refere-se geralmente a polinucleotídeos curtos e de fita simples, geralmente sintéticos, que normalmente apresentam, mas não necessariamente, menos de cerca de 200 nucleotídeos de comprimento. Os termos “oligonucleotídeo” e "polinucleotídeo" não são mutuamente exclusivos. A descrição acima para polinucleotídeos é igualmente e completamente aplicável para oligonucleotídeos.
[184] “Percentual (%) de identidade de sequência de aminoácidos” no presente é definido como o percentual de resíduos de aminoácidos na possível sequência que é idêntico aos resíduos de aminoácidos na sequência do polipeptídeos ou peptídeo específico, após o alinhamento das sequências e introdução de intervalos, se necessário, para atingir o percentual máximo de identidade de sequências, sem considerar nenhuma substituição conservadora como parte da identidade de sequências. O alinhamento para propósitos da determinação do percentual de identidade de sequências de aminoácidos pode ser atingido de várias formas que se encontram dentro do conhecimento da técnica, tais como o uso de softwares de computador disponíveis ao público, tal como os softwares BLAST, BLAST-2, ALIGN, ALIGN- 2 ou Megalign (DNASTAR). Os técnicos no assunto podem determinar parâmetros apropriados para medir o alinhamento, incluindo qualquer algoritmo necessário para atingir o alinhamento máximo ao longo do comprimento total das sequências que estão sendo comparadas. Para os propósitos do presente, entretanto, a % de identidade de sequências de aminoácidos é gerada utilizando o programa de computador de comparação de sequências ALIGN-2, em que o código fonte completo do programa ALIGN-2 é fornecido na tabela A abaixo. O programa de computador de comparação de sequências ALIGN-2 é de autoria da Genentech, Inc., e o código fonte foram depositados com a documentação de usuário no Escritório Norte-Americano de Direitos Autorais, Washington DC 20559, Estados Unidos, onde foi registrado com o n° de Registro de Direitos Autorais: TXU510087. O programa ALIGN-2 está publicamente disponível pela Genentech, Inc., South San Francisco, Califórnia, ou pode ser compilado a partir do código-fonte fornecido, por exemplo, na publicação WO2007/001851. O programa ALIGN-2 deve ser compilado para uso em um sistema operacional UNIX, preferencialmente o UNIX digital V4.0D. Todos os parâmetros de comparação de sequência são definidos pelo programa ALIGN-2 e não variam.
[185] Em situações onde o ALIGN-2 é empregado para a comparação de sequências de aminoácidos, a % de identidade de sequência de aminoácidos de uma dada sequência de aminoácidos A, com, ou contra uma dada sequência de aminoácidos B (que pode alternativamente ser formulada como uma determinado sequência de aminoácido A que contenha uma certa % de identidade de sequência de aminoácidos com, ou contra uma dada sequência de aminoácidos B) é calculado da seguinte forma:100 vezes a fração X/Y
[186] no qual, X é a quantidade de resíduos de aminoácidos avaliados como coincidências idênticas pelo programa de alinhamento de sequências ALIGN-2 no alinhamento de A e B naquele programa e em que Y é a quantidade total de resíduos de aminoácidos em B. Apreciar-se-á que, quando o comprimento da sequência de aminoácidos A não for igual ao comprimento da sequência de aminoácidos B, o percentual de identidade de sequências de aminoácidos de A para B não seja igual ao percentual de identidade de sequências de aminoácidos de B para A.
[187] Em alguns exemplos de realização, duas ou mais sequências de aminoácidos são pelo menos 50%, 60%, 70%, 80%, ou 90% idênticas. Em alguns exemplos de realização, duas ou mais sequências de aminoácidos são pelo menos 95%, 97%, 98%, 99%, ou 100% idênticas. A menos que especificamente indicado em contrário, todos os valores percentuais de identidade de sequências de aminoácidos utilizados no presente são obtidos, tal como descrito no parágrafo anterior, pelo uso do programa ALIGN-2.
[188] O termo "FGFR3", da forma como é utilizado no presente, refere-se, a menos que especificado ou indicado de outra forma, a qualquer polipeptídeo nativo ou variante (seja natural ou sintético) de FGFR3 (por exemplo, isoforma FGFR3-IIIb ou isoforma FGFR3-IIIc). O termo "sequência nativa" abrange especificamente aquelas que ocorrem naturalmente sob a forma truncada ou secretada (por exemplo, uma sequência do domínio extracelular ou uma sequência da subunidade transmembrana), formas variantes que ocorrem naturalmente (por exemplo, formas com splicing alternativo) e variantes alélicos que ocorrem naturalmente. O termo “FGFR3 tipo selvagem” refere-se em geral a um polipeptídeo que compreende a sequência de aminoácidos de uma proteína FGFR3 de ocorrência natural. A Expressão “sequência de FGFR3 tipo selvagem” refere-se em geral a uma sequência de aminoácidos que é encontrada no FGFR3 de ocorrência natural.
[189] O termo “ligante de FGFR3”, (alternadamente denominado “FGF”), da forma como é utilizado no presente documento, salvo quando indicado de outra maneira ou contextualmente, refere-se a qualquer polipeptídeo ligante de FGFR3 nativo ou variante (natural ou sintético) (por exemplo, FGF1, FGF2, FGF4, FGF8, FGF9, FGF17, FGF18, FGF23). O termo "sequência nativa" abrange especificamente aquelas que ocorrem naturalmente sob a forma truncada ou secretada (por exemplo, uma sequência do domínio extracelular ou uma sequência da subunidade transmembrana), formas variantes que ocorrem naturalmente (por exemplo, formas com splicing alternativo) e variantes alélicos que ocorrem naturalmente. O termo “ligante de FGFR3 tipo selvagem” refere-se em geral a um polipeptídeo que compreende a sequência de aminoácidos de uma proteína ligante de FGFR3 de ocorrência natural. A Expressão “sequência de ligante de FGFR3 tipo selvagem” refere-se em geral a uma sequência de aminoácidos que é encontrada no ligante de FGFR3 de ocorrência natural.
[190] A “ativação de FGFR3” refere-se à ativação ou fosforilação do receptor FGFR3. Geralmente, a ativação de FGFR3 resulta na transdução de sinal (por exemplo, que é causada por um domínio quinase intracelular do receptor FGFR3 que fosforila resíduos de tirosina no FGFR3 ou um polipeptídeo substrato). A ativação FGFR3 pode ser mensurada pela ligação do ligante de FGFR a um receptor FGFR3 de interesse. A ligação de um ligante de FGFR3 (por exemplo, FGF1 ou FGF9) ao FGFR3 pode ativar um domínio quinase do FGFR3 e, assim, resultar na fosforilação de resíduos de tirosina no FGFR3 e/ou a fosforilação de resíduos de tirosina em polipeptídeo(s) substrato adicional(s).
[191] O termo "constitutivo", conforme utilizado no presente, por exemplo, aplicado a atividade quinase, refere-se à atividade de sinalização continua de um receptor que não é dependente da presença de um ligante ou outra molécula ativadora. Dependendo da natureza do receptor, toda a atividade pode ser constitutiva ou a atividade do receptor pode ser ainda mais ativada por meio da ligação de outras moléculas (por exemplo, ligantes). Eventos celulares que levam à ativação de receptores são bem conhecidos entre os técnicos do assunto. Por exemplo, a ativação pode incluir uma oligomerização, por exemplo, dimerização, trimerização, etc., em complexos receptores de ordem maior. Complexos podem incluir uma única espécie de proteína, ou seja, um complexo homomérico. Alternativamente, os complexos podem incluir pelo menos duas espécies diferentes de proteínas, ou seja, um complexo heterodimérico. A formação do complexo pode ser causada, por exemplo, pela superexpressão de formas normais ou mutantes de receptor na superfície de uma célula. A formação do complexo pode também ser causada por uma mutação específica ou mutações em um receptor.
[192] O termo "independente de ligante", conforme utilizado no presente, por exemplo, aplicado a atividade de sinalização do receptor, refere- se à atividade de sinalização que não é dependente da presença de um ligante. Um receptor que possui atividade quinase independente de ligante não exclui necessariamente a ligação do ligante ao receptor para produzir uma ativação adicional da atividade de quinase.
[193] O termo "dependente de ligante", conforme utilizado no presente, por exemplo, aplicado a atividade de sinalização do receptor, refere- se à atividade de sinalização que é dependente da presença de um ligante.
[194] A frase “amplificação do gene” refere-se a um processo pelo qual as múltiplas cópias de um gene ou de um fragmento do gene são formadas em uma célula ou em uma linhagem de célula particular. A região duplicada (um estiramento do DNA amplificado) é frequentemente referida como “amplicon”. Geralmente, a quantidade de RNA mensageiro (mRNA) produzido, ou seja, o nível de expressão gênica, também aumenta na proporção do número de cópias feitas do gene particular expresso.
[195] Um “inibidor de tirosina quinase” é uma molécula que inibe a atividade tirosina quinase de uma tirosina quinase, tal como de um receptor FGFR3.
[196] Um câncer ou amostra biológica que “exibe expressão, amplificação ou ativação de FGFR3” é aquele que, em um teste diagnóstico, expressa (incluindo a superexpressão) FGFR3, têm o gene FGFR3 amplificado, e/ou demonstra de outra forma a ativação ou fosforilação de um FGFR3.
[197] Um câncer ou amostra biológica que “exibe ativação de FGFR3” é aquele que, em um teste de diagnóstico, demonstra ativação ou fosforilação de FGFR3. Essa ativação pode ser determinada diretamente (por exemplo, mensurando a fosforilação de FGFR3 por ELISA) ou indiretamente.
[198] Um câncer ou amostra biológica que “exibe ativação constitutiva de FGFR3” é aquele que, em um teste de diagnóstico, demonstra ativação ou fosforilação constitutiva de um FGFR3. Essa ativação pode ser determinada diretamente (por exemplo, mensurando a fosforilação de c-FGFR3 por ELISA) ou indiretamente.
[199] Um câncer ou amostra biológica que “exibe amplificação de FGFR3” é aquele que, em um teste de diagnóstico, possui o gene FGFR3 amplificado.
[200] Um câncer ou amostra biológica que “exibe translocação de FGFR3” é aquele que, em um teste de diagnóstico, possui o gene FGFR3 traslocado. Um exemplo de uma translocação de FGFR3 é a translocação t(4, 14), que ocorre em alguns tumores de mieloma.
[201] Um “ensaio fosfo-ELISA” é no presente um ensaio em que a fosforilação de um ou mais FGFR3, substrato ou molécula de sinalização a jusante (downstream) é(são) avaliado(s) por um ensaio imunoadsorvente enzima-associado (ELISA) utilizando um reagente, normalmente um anticorpo para detectar o FGFR3, substrato ou molécula de sinalização a jusante. Em alguns exemplos de realização, é usado um anticorpo que detecta FGFR3 fosforilado ou pMAPK fosforilado. O ensaio pode ser executado em lisados de células, preferivelmente em amostras biológicas frescas ou congeladas.
[202] Um câncer ou amostra biológica que “exibe ativação de FGFR3 independente de ligante” é aquele que, em um teste de diagnóstico, demonstra ativação ou fosforilação de FGFR3 de maneira independente de ligante. Essa ativação pode ser determinada diretamente (por exemplo, mensurando a fosforilação de FGFR3 por ELISA) ou indiretamente.
[203] Um câncer ou amostra biológica que “exibe ativação de FGFR3 dependente de ligante” é aquele que, em um teste de diagnóstico, demonstra ativação ou fosforilação de FGFR3 de maneira dependente de ligante. Essa ativação pode ser determinada diretamente (por exemplo, mensurando a fosforilação de FGFR3 por ELISA) ou indiretamente.
[204] Um câncer ou amostra biológica que “exibe ativação de FGFR3 independente de ligante” é aquele que, em um teste de diagnóstico, demonstra ativação ou fosforilação de um FGFR3 de maneira independente de ligante. Essa ativação pode ser determinada diretamente (por exemplo, mensurando a fosforilação de FGFR3 por ELISA) ou indiretamente.
[205] Uma célula de câncer com “superexpressão ou amplificação de FGFR3” é aquela que apresenta níveis significativamente mais elevados de proteína ou gene FGFR3 quando comparada com uma célula não cancerosa do mesmo tipo de tecido. Tal superexpressão pode ser causada pela amplificação do gene ou por um aumento da transcrição ou tradução. A superexpressão ou amplificação de FGFR3 pode ser determinada em um ensaio diagnóstico ou prognóstico, pela avaliação de níveis aumentados da proteína FGFR# presente na superfície de uma célula (por exemplo, por meio de um ensaio de imunohistoquímica; IHQ). Alternativamente, ou adicionalmente, podem ser mensurados os níveis de ácido nucleico que codificam FGFR3 na célula, por exemplo, por meio da hibridização in situ fluorescente (FISH; vide, publicação WO 98/45479 publicado em outubro de 1998), técnicas de Southern blotting, ou reação em cadeia da polimerase (PCR), como PCR quantitativo em tempo real (qRT-PCR). Com exceção dos ensaios acima, vários ensaios in vivo e ex vivo estão disponíveis aos profissionais hábeis no assunto. Por exemplo, um ensaio que possa expor célula do corpo do paciente a um anticorpo que é opcionalmente marcado com um marcador detectável, por exemplo, um isótopo radioativo, e a ligação do anticorpo com as células do paciente pode ser avaliada, por exemplo, por varredura externa da radioatividade ou pela análise de uma biópsia retirada de um paciente que foi exposto previamente ao anticorpo.
[206] O termo “mutação”, conforme utilizado no presente, significa uma diferença na sequência de ácidos nucleicos ou aminoácidos de um determinado ácido nucleico (gene, RNA) ou proteína em relação ao ácido nucleico ou proteína do tipo selvagem, respectivamente. Uma proteína ou ácido nucleico mutante pode ser expresso a partir de, ou encontrado em um alelo (heterozigose) ou ambos os alelos (homozigose) de um gene, e pode ser somática ou germinal. Na presente invenção, as mutações são geralmente somáticas. Mutações incluem rearranjos de sequência, como inserções, deleções e mutações pontuais (incluindo de um único nucleotídeo/polimorfismos de aminoácidos).
[207] "Inibir" é diminuir ou reduzir uma atividade, função e/ou quantidade em comparação com uma referência.
[208] Um agente possui “atividade ou função agonista” quando um agente mimetiza pelo menos uma das atividades funcionais de um polipeptídeo de interesse (por exemplo, ligante de FGFR, como FGF1 ou FGF9).
[209] Um “anticorpo agonista”, conforme utilizado no presente, é um anticorpo que mimetiza pelo menos uma das atividades funcionais de um polipeptídeo de interesse (por exemplo, ligante de FGFR, como FGF1 ou FGF9).
[210] “Expressão” da proteína refere-se a conversão da informação codificada em um gene para RNA mensageiro (mRNA) e depois em uma proteína.
[211] No presente, uma amostra ou célula que “expresse” uma proteína de interesse (tal como um receptor FGF ou ligante do receptor FGF) é aquela na qual o mRNA que codifica a proteína, ou a própria proteína, incluindo fragmentos desta, é determinada por estar presente na amostra ou célula.
[212] Um “imunoconjugado” (designado de maneira intercambiável por “conjugado droga-anticorpo” ou “ADC”) significa um anticorpo conjugado a um ou mais agentes citotóxicos, tais como um agente quimioterápico, um medicamento, um agente inibidor do crescimento, uma toxina (por exemplo, uma toxina de proteína, uma toxina enzimaticamente ativa de origem bacteriana, vegetal, fúngica ou animal, ou fragmentos destas), ou um isótopo radioativo (ou seja, um radioconjugado).
[213] O termo "região Fc", da forma como utilizado no presente, se refere em geral a um complexo que compreende as sequências polipeptídicas da extremidade C-terminal de uma cadeia pesada de imunoglobulina, onde uma sequência polipeptídica C-terminal é aquela que é obtida por digestão de um anticorpo intacto pela papaína. A região de Fc pode ser uma sequência nativa ou variante da região Fc. Embora os limites da sequência Fc da cadeia pesada de uma imunoglobulina possam variar, a sequência Fc da cadeia pesada da IgG humana é normalmente definida como se estendendo a partir de um resíduo de aminoácidos por volta da posição Cys226, ou por volta da posição Pro230, até a região carboxi-terminal da sequência Fc. A sequência Fc de uma imunoglobulina compreende geralmente dois domínios constantes, um domínio CH2 e um domínio CH3, e compreende opcionalmente um domínio CH4. A lisina C-terminal (resíduo 477 de acordo com sistema de numeração EU) da região Fc pode ser removida, por exemplo, durante a purificação do polipeptídeo, ou pela construção de um ácido nucleico recombinante codificador do anticorpo. Consequentemente, uma composição compreendendo um anticorpo contendo uma região Fc de acordo com a presente invenção, pode compreender com K447, com todos os resíduos K447 removidos, ou uma mistura de anticorpos com e sem resíduos K447.
[214] A expressão “polipeptídeo Fc” significa no presente um dos polipeptídeos que compõem uma região Fc. Um polipeptídeo Fc pode ser obtido a partir de qualquer imunoglobulina adequada, tal como os subtipos IgG1, IgG2, IgG3 ou IgG4, IgA, IgE, IgD ou IgM. Em algumas realizações, um polipeptídeo Fc compreende uma parte ou a totalidade de uma sequência de articulação (hinge) tipo selvagem (geralmente em sua extremidade N-terminal). Em algumas realizações, um polipeptídeo Fc não compreende uma sequência de articulação funcional ou tipo selvagem.
[215] Um anticorpo de “bloqueio” ou anticorpo “antagonista” é aquele que inibe ou reduz a atividade biológica do antígeno ao qual se liga. Anticorpos de bloqueio ou anticorpos antagonistas preferidos inibem de forma completa a atividade biológica do antígeno.
[216] Um “anticorpo nu” é um anticorpo não conjugado a uma molécula heteróloga, tal como uma fração citotóxica ou um radiomarcador.
[217] Um anticorpo possuindo uma “característica biológica” de um determinado anticorpo é aquele que possui uma ou mais características biológicas do qual os anticorpos que o distinguem de outros anticorpos se ligam ao mesmo antígeno.
[218] Com o intuito de selecionar anticorpos que se ligam a um epítopo sobre um antígeno ligado a um anticorpo de interesse, pode ser executado um ensaio de bloqueio cruzado rotineiro, tal como aquele descrito em Antibodies, A Laboratory Manual, Cold Spring Harbor Laboratory, Ed. Harlow e David Lane (1988).
[219] Para aumentar a meia vida do anticorpo ou polipeptídeo contendo a sequência de aminoácidos da presente invenção, pode-se incorporar um epítopo ligado ao receptor de recuperação do anticorpo (especialmente um fragmento de anticorpo), conforme descrito, por exemplo, na patente US 5.739.277. Por exemplo, uma molécula de ácido nucleico que codifica o receptor de recuperação do epítopo de ligação pode estar ligada no quadro de leitura a um ácido nucleico que codifica uma sequência de polipeptídeo da presente invenção de modo que a proteína de fusão expressa pela molécula de ácido nucleico desenvolvida por engenharia genética compreende o receptor de recuperação do epítopo de ligação e uma sequência polipeptídica da presente invenção. Da forma como utilizada no presente, a expressão “epítopo de ligação ao receptor de recuperação” designa um epítopo da região Fc de uma molécula de IgG (por exemplo, IgG1, IgG2, IgG3 ou IgG4) que é responsável pelo aumento da meia vida in vivo no soro da molécula de IgG (por exemplo, Ghetie et al., Ann. Rev. Immunol. 18:739-766 (2000), Tabela 1). Anticorpos com substituições em uma região Fc deste e aumento na meia-vida sérica também estão descritos nos documentos WO 00/42072, WO 02/060919; e em Shields et al. J. Biol. Chem. 276:6591-6604 (2001); Hinton, J. Biol. Chem. 279:6213-6216 (2004). Em outra realização, a meia-vida sérica também pode ser aumentada, por exemplo, anexando outras sequências de polipeptídeos. Por exemplo, anticorpos ou outros polipeptídeos úteis nos métodos da presente invenção podem ser anexados à albumina do soro ou uma porção da albumina do soro que se liga aos receptores FcRn ou um peptídeo que se liga a albumina, para que albumina do soro se ligue ao anticorpo ou polipeptídeo, por exemplo, as sequências polipeptídicas divulgadas na publicação WO 01/45746. Em um exemplo de realização, o peptídeo da albumina do soro a ser acoplado compreende a sequência de aminoácidos DICLPRWGCLW (SEQ ID No: 183). Em outro exemplo de realização, a meia-vida de um Fab é aumentada por estes métodos. Vide também, Dennis, M. S., et al., J. Biol. Chem. 277(38):35035-35043 (2002) para sequências de peptídeo que se ligam a albumina.
[220] “Fragmento” significa uma parte de um polipeptídeo ou molécula de ácido nucleico que contém, preferencialmente, pelo menos 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95% ou mais de todo ocomprimento da molécula de ácido nucleico ou polipeptídeo de referência. Um fragmento pode conter 10, 20, 30, 40, 50, 60, 70, 80, 90 ou 100, 200, 300, 400, 500, 600, ou mais nucleotídeos ou 10, 20, 30, 40, 50, 60, 70, 80, 90, 100, 120, 140, 160, 180, 190, 200 ou mais aminoácidos.
[221] A frase “pouca ou nenhuma função agonista” com relação a um anticorpo da presente invenção, conforme utilizado no presente, significa que o anticorpo não provoca uma quantidade de atividade agonista biologicamente significativa, por exemplo, pela administração do anticorpo a um paciente. Como seria compreendido no estado da técnica, a quantidade de uma atividade pode ser determinada quantitativamente e qualitativamente, desde que possa ser feita uma comparação entre um anticorpo da presente invenção e uma contraparte de referência. A atividade pode ser mensurada ou detectada de acordo com qualquer ensaio ou técnica conhecida no estado da técnica, incluindo, por exemplo, aquelas descritas na presente invenção. A quantidade de atividade de um anticorpo da presente invenção e de seu homólogo de referência pode ser determinada por ensaios paralelos ou separados. Em alguns exemplos de realização, um anticorpo bivalente da presente invenção não possui função agonista substancial.
[222] Os termos “apoptose” e “atividade apoptótica” são usados no sentido mais amplo e referem-se a forma ordenada ou controlada de morte celular que ocorre nos mamíferos e que normalmente é acompanhada por uma ou mais alterações celulares características, incluindo a condensação citoplasmática, perda das microvilosidades da membrana plasmática, segmentação do núcleo, degradação do DNA cromossômico ou perda de função mitocondrial. Esta atividade pode ser determinada e mensurada usando técnicas conhecidas, por exemplo, por meio de ensaios da viabilidade celular, análise por FACS ou eletroforese de DNA, e mais especificamente pela ligação da anexina V, fragmentação do DNA, retração celular, dilatação do retículo endoplasmático, fragmentação da célula, e/ou formação de vesículas na membrana (denominadas de corpos apoptóticos).
[223] Os termos “anticorpo” e “imunoglobulina” são utilizados de forma alternada e no sentido mais amplo e incluem anticorpos monoclonais (por exemplo, anticorpos monoclonais de comprimento total ou intactos), anticorpos policlonais, anticorpos multivalentes, anticorpos multiespecíficos (por exemplo, anticorpos biespecíficos contanto que eles apresentem a atividade biológica desejada), e podem incluir ainda determinados fragmentos de anticorpos (conforme descrito em maiores detalhes na presente invenção).
[224] Um anticorpo pode ser humano, humanizado e/ou maturado por afinidade.
[225] O termo "variável" refere-se ao fato de que certas partes dos domínios variáveis diferem amplamente na sequência entre os anticorpos e são utilizados na especificidade de ligação de cada anticorpo específico para o seu antígeno específico. No entanto, a variabilidade não é distribuída uniformemente ao longo de todos os domínios variáveis de anticorpos. Ela é tipicamente concentrada em três segmentos denominados regiões determinantes de complementaridade (CDRs) ou regiões hipervariáveis, tanto nos domínios variáveis de cadeia leve como de cadeia pesada. As porções mais bem conservadas de domínios variáveis são denominadas de regiões estruturais (ou de arcabouço ou frameworks) (FR). Os domínios variáveis de cadeias leves e pesadas nativas compreendem 4 regiões de FRs, adotando em grande parte uma configuração em folha-β, conectadas por 3 CDRs, que formam conexões em alças (loops) e, em alguns casos, fazem parte da estrutura em folha-β. As CDRs de cada cadeia são mantidas juntas em grande proximidade pelas regiões FR e com as CDRs da outra cadeia, contribuindo para a formação do sítio de ligação ao antígeno dos anticorpos (vide Kabat et al., Sequences of Proteins of Immunological Interest, Quinta Edição, Public Health Service, National Institutes of Health, Bethesda, MD, (1991)). Os domínios constantes não estão envolvidos diretamente na ligação de um anticorpo a um antígeno, mas exibem várias funções efetoras, tais como a participação do anticorpo na toxicidade celular dependente de anticorpos.
[226] A digestão de anticorpos pela papaína produz dois fragmentos idênticos de ligação ao antígeno, denominados fragmentos “Fab”, cada qual com um único sítio de ligação ao antígeno, e um fragmento “Fc” residual, cujo nome reflete a sua capacidade de rápida cristalização. O tratamento com a pepsina gera um fragmento F(ab’)2 que contém dois sítios de ligação a antígenos e ainda é capaz de reticular o antígeno.
[227] “Fv” é o menor fragmento de anticorpo, que contém um sítio completo de reconhecimento e de ligação de antígenos. Em uma cadeia de duas espécies de Fv, esta região consiste em um dímero de um domínio variável de cadeia pesada e em um de cadeia leve em estreita associação não covalente. Em uma única cadeia de Fv, um domínio variável da cadeia pesada e um da cadeia leve podem ser covalentemente ligados por um peptídeo ligante flexível de tal forma que a cadeias leves e pesadas possam se associar em uma estrutura "dimérica" análoga àquela do Fv de duas cadeias. É nesta configuração que as três CDRs de cada domínio variável interagem para definir um sítio de ligação aos antígenos sobre a superfície do dímero VH-VL. Coletivamente, as seis CDRs conferem a especificidade de ligação para o anticorpo. Entretanto, mesmo um único domínio variável (ou metade de um Fv que compreende apenas três CDRs específicos para um antígeno), possui a capacidade de reconhecer e ligar ao antígeno, embora em menor afinidade do que o site de ligação inteiro.
[228] O fragmento Fab também contém o domínio constante da cadeia leve e o primeiro domínio constante (CH1) da cadeia pesada. Os fragmentos Fab’ diferem dos fragmentos Fab pela adição de alguns resíduos no carbóxi-terminal do domínio CH1 de cadeia pesada, incluindo uma ou mais cisteínas da região de articulação do anticorpo. Fab’-SH é a designação da presente invenção para o Fab’ em que o(s) resíduo(s) de cisteína dos domínios constantes sustenta(m) um grupo tiol livre. Os fragmentos de anticorpos F(ab’)2 foram produzidos originalmente como pares de fragmentos Fab’ que possuem dobradiças de cisteínas entre si. Outros acoplamentos químicos de fragmentos de anticorpos são também conhecidos.
[229] As “cadeias leves” de anticorpos (imunoglobulinas) de qualquer espécie de vertebrado podem ser atribuídas a um dentre dois tipos claramente distintos, denominados de kappa (K) e lambda (À), com base nas sequências de aminoácidos de seus domínios constantes.
[230] Dependendo da sequência de aminoácidos do domínio constante das suas cadeias pesadas, os anticorpos podem ser atribuídos a diferentes classes. Existem cinco classes principais de imunoglobulinas: IgA, IgD, IgE, IgG e IgM, e várias delas podem ser divididas em subclasses (isótipos), por exemplo, IgG1, IgG2, IgG3, IgG4, IgA1 e IgA2. Os domínios constantes de cadeia pesada que correspondam às diferentes classes de imunoglobulinas são denominados de α, δ, ε, Y, e μ, respectivamente. As estruturas de subunidade e as configurações tridimensionais das diferentes classes de imunoglobulinas são bem conhecidas. “Fragmentos de anticorpos” compreendem uma porção de anticorpo intacto, em que a porção retém preferencialmente pelo menos um, e preferencialmente a maioria ou todas as funções normalmente associadas a essa porção quando presente em um anticorpo intacto. Exemplos de fragmentos de anticorpos incluem Fab, Fab’, F(ab’)2 e fragmentos Fv, diacorpos, anticorpos lineares, moléculas de anticorpos de cadeia única e anticorpos multiespecíficos formados a partir de fragmentos de anticorpos. Em uma realização, o fragmento de anticorpo compreende de um sítio de ligação ao antígeno do anticorpo intacto mantendo assim a capacidade de se ligar ao antígeno. Em outra realização, um fragmento de anticorpo, por exemplo, um que compreende a região Fc, retém ao menos uma das funções biológicas normalmente associadas com a região Fc quando presente em um anticorpo intacto, tal como a ligação de FcRn, modulação da meia vida do anticorpo, função ADCC e ligação do complemento. Em um exemplo de realização, um fragmento de anticorpo é um anticorpo monovalente que possui uma meia vida in vivo substancialmente similar ao de um anticorpo intacto. Por exemplo, tal fragmento de anticorpo pode compreender de um braço de ligação ao antígeno para uma sequência Fc capaz de conferir estabilidade in vivo ao fragmento.
[231] O termo “região hipervariável”, “HVR”, ou “HV”, quando utilizado no presente refere-se às regiões de um domínio variável de anticorpo que são hipervariáveis na sequência e/ou formam alças estruturalmente definidas. Geralmente, os anticorpos compreendem seis regiões hipervariáveis; sendo três na VH (H1, H2, H3), e três na VL (L1, L2, L3). Diversos delineamentos da região hipervariável são utilizados e englobados neste documento. As regiões determinantes de complementaridade de Kabat (CDRs) são baseadas na variabilidade das sequências e são as mais comumente usadas (Kabat et al. Sequences of Proteins of Immunological Interest, 5a Ed, National Institute of Health, Bethesda, Md. 1991). Chothia por sua vez refere-se à localização das alças estruturais (Chothia e Lesk J. Mol. Biol. 196:901-917 (1987)). As regiões hipervariável do AbM representam um acordo entre os CDRs de Kabat e as alças (loops) estruturais de Chothia, e são utilizados pelo programa “Oxford Molecular’s AbM antibody modeling software”. O "contato" das regiões hipervariáveis é baseado em uma análise das estruturas de complexo cristalino disponíveis. Os resíduos de cada uma destas regiões hipervariáveis são descritas a seguir.Alça Kabat AbM Chothia ContatoL1 L24-L34 L24-L34 L26-L32 L30-L36L2 L50-L56 L50-L56 L50-L52 L46-L55L3 L89-L97 L89-L97 L91-L96 L89-L96H1 H31-H35B H26-H35B H26-H32 H30-H35B(Numeração de Kabat)H1 H31-H35 H26-H35 H26-H32 H30-H35 (Numeração de Chothia)H2 H50-H65 H50-H58 H53-H55 H47-H58H3 H95-H102 H95-H102 H96-H101 H93-H101
[232] As regiões hipervariáveis podem compreender "regiões hipervariáveis alongadas", como as seguintes: 24-36 ou 24-34 (L1), 46-56 ou 5056 (L2) e 89-97 (L3) no VL e 26-35 (H1), 50-65 ou 49-65 (H2) e 93-102, 94-102 ou 95-102 (H3) no VH. Os resíduos do domínio variável são numerados de acordo com a Kabat et al., supra para cada uma dessas definições.
[233] Resíduos da "região estrutural" (framework) ou "FR" são aqueles resíduos do domínio variável que não são resíduos da região hipervariável conforme definido no presente.
[234] Formas “humanizadas” de anticorpos não humanos (tal como, murino) são anticorpos quiméricos que contém uma sequência mínima derivada de uma imunoglobulina não humana. Na maior parte, os anticorpos humanizados são imunoglobulinas humanas (anticorpo receptor) em que os resíduos de uma região hipervariável do receptor são substituídos por resíduos de uma região hipervariável de uma espécie não humana (anticorpo doador) tal como camundongo, rato, coelho ou primata não humano possuindo a especificidade, afinidade e/ou capacidade desejada(s). Em alguns casos, resíduos da região estrutural (FR) da imunoglobulina humana são substituídos por resíduos não humanos correspondentes. Adicionalmente, anticorpos humanizados podem conter resíduos que não são encontrados no anticorpo receptor ou no anticorpo doador. Estas modificações podem ser feitas para refinar o desempenho do anticorpo. Em geral, o anticorpo humanizado compreenderá substancialmente todos de pelo menos um, e tipicamente dois, domínios variáveis, em que todas ou substancialmente todas das alças (loops) hipervariáveis correspondem aos de uma imunoglobulina não humana e todas ou substancialmente todas as FRs são de uma sequência de imunoglobulina humana. O anticorpo humanizado poderá também compreender opcionalmente pelo menos uma porção de uma região constante de imunoglobulina (Fc), tipicamente a de uma imunoglobulina humana. Para maiores detalhes, vide Jones et al., Nature 321:522-525 (1986); Riechmann et al., Nature 332:323-329 (1988); e Presta, Curr. Op. Struct. Biol. 2:593-596 (1992). Veja também os artigos de revisão e as referências citadas nos mesmos: Vaswani e Hamilton, Ann. Allergy, Asthma & Immunol. 1:105-115 (1998); Harris, Biochem. Soc.Transactions 23:1035-1038 (1995); Hurle e Gross, Curr. Op. Biotech. 5:428-433 (1994).
[235] Os anticorpos (imunoglobulinas) “quiméricos” possuem uma porção da cadeia leve e/ou pesada que seja idêntica, ou homóloga, com as sequências correspondentes em anticorpos derivados de uma espécie especifica, ou pertencem a uma classe ou subclasse específica de anticorpo, enquanto o restante da cadeia é idêntico, ou homólogo, com as sequências correspondentes em anticorpos derivados de outras espécies, ou que pertencem a outra classe ou subclasse de anticorpo, bem como fragmentos destes anticorpos, desde que eles exibam a atividade biológica desejada (Patente US 4.816.567; e Morrison et al., Proc. Natl. Acad. Sci. USA 81:6851-6855 (1984)). Um anticorpo humanizado, conforme utilizado na presente invenção é um subconjunto de anticorpos quiméricos.
[236] Fragmentos de anticorpo “Fv de cadeia única” ou “scFv” compreendem os domínios VH e VL de anticorpo, no qual estes domínios estão presentes em uma única cadeia de polipeptídeo. Geralmente, o polipeptídeo scFv compreende ainda um polipeptídeo ligante entre os domínios VH e VL, permitindo que o scFv forme a estrutura desejada para ligação ao antígeno. Para uma revisão de scFv vide Pluckthun, em The Pharmacology of Monoclonal Antibodies, vol. 113, Eds Rosenburg e Moore., Springer-Verlag, Nova Iorque, págs. 269-315 (1994).
[237] Um “antígeno” é um antígeno predeterminado ao qual um anticorpo pode se ligar seletivamente. O antígeno alvo pode ser polipeptídeo, carboidratos, ácidos nucleicos, lipídio, ou outros haptenos de ocorrência natural ou composto sintético. Preferivelmente, o antígeno alvo é um polipeptídeo.
[238] O termo “diacorpos” refere-se a pequenos fragmentos de anticorpos com dois sítios de ligação ao antígeno, em que tais fragmentos compreendem um domínio variável de cadeia pesada (VH) conectado a um domínio variável de cadeia leve (VL) na mesma cadeia polipeptídica (VH - VL). Por meio do uso de um ligante que é muito curto para permitir o pareamento entre os dois domínios na mesma cadeia, os domínios são forçados a parear com os domínios complementares de outra cadeia, e criar dois sítios de ligação ao antígeno. Diacorpos estão descritos em maiores detalhes, por exemplo, na Patente EP 404.097; publicação WO 93/11161; e Hollinger et al., Proc. Natl. Acad. Sci. USA 90: 6444-6448 (1993).
[239] Um “anticorpo humano” é aquele que possui uma sequência de aminoácido que corresponde à sequência de um anticorpo produzido por um humano e/ou foi produzido pelo uso de qualquer técnica para a produção de anticorpos humanos conforme descrito no presente. Esta definição de um anticorpo humano exclui especificamente um anticorpo humanizado, que compreende resíduos não humanos de ligação ao antígeno.
[240] Um anticorpo “maturado por afinidade” é um anticorpo que possui uma ou mais alterações em um ou mais CDRs do mesmo que resulta na melhora da afinidade de ligação do anticorpo ao antígeno, comparado ao anticorpo parental que não possui tal(ais) alteração(ões). Os anticorpos maturados por afinidade preferidos possuirão afinidades nanomolares ou até picomolares para o antígeno alvo. Anticorpos maturados por afinidade são produzidos por procedimentos conhecidos no estado da técnica. Marks, et al., Biotechnology 10:779-783 (1992) descrevem a maturação por afinidade pelo método de shuffling do domínio VL e VH. A mutagênese aleatória de CDR e/ou resíduos da região de estrutural (ou de arcabouço (framework)) é descrita por: Barbas et al., Proc Nat. Acad. Sci. USA 91:3809-3813 (1994); Schier et al., Gene 169:147-155 (1995); Yelton et al., J. Immunol. 155:1994-2004 (1995); Jackson et al., J. Immunol. 154(7):3310-9 (1995); e Hawkins et al., J. Mol. Biol. 226:889896 (1992).
[241] Um anticorpo com "funções efetoras" refere-se as atividades biológicas atribuídas a região Fc (uma sequência nativa da região Fc ou uma sequência de aminoácido variante da região Fc) de um anticorpo, e variam de acordo com o isotipo do anticorpo. Exemplos de funções efetoras de anticorpos incluem: ligação de C1q e citotoxicidade dependente de complemento; ligação de receptor Fc; citotoxicidade mediada por célula dependente de anticorpo (ADCC); fagocitose; regulação negativa dos receptores de superfície celular (por exemplo, receptor de célula B), e ativação de células B.
[242] “Citotoxicidade mediada por células dependente de anticorpo” e “ADCC” referem-se a uma forma de citotoxicidade em que a Ig secretada ligada aos receptores Fc (FcRs) presentes em certos tipos de células citotóxicas (por exemplo, células Matadoras Naturais (natural killer - NK), neutrófilos e macrófagos) permitem que estas células efetoras citotóxicas se liguem especificamente a um antígeno sobre a célula-alvo e, em seguida, mate a célula-alvo com citotoxinas. Os anticorpos "armam" as células citotóxicas e são absolutamente necessários para tal lise. As células primárias para a mediação de ADCC, as células NK, expressam unicamente FcyRIII, enquanto monócitos expressam FcyRI, FcyRII e FcyRIII. A expressão FcR em células hematopoiéticas é resumida na tabela 3, página 464 de Ravetch et al., Annu. Rev. Immunol. 9:457-92 (1991). Para avaliar a atividade ADCC de uma molécula de interesse, podem ser realizados ensaios ADCC in vitro, tais como os descritos nas Patentes US 5.500.362 ou US 5.821.337 ou Patente US 6.737.056 de Presta. As células efetoras úteis para tais testes incluem células mononucleares de sangue periférico (PBMC) e células natural killer (NK). Alternativamente ou adicionalmente, a atividade ADCC da molécula de interesse pode ser avaliada in vivo, por exemplo, em um modelo animal tal como descrito em Clynes et al., PNAS (USA) 95:652-656 (1998).
[243] "Células efetoras humanas” são leucócitos que expressam uma ou mais FcRs e realizam funções efetoras. Preferencialmente, as células expressam pelo menos FcyRIII e realizam função efetora de ADCC. Exemplos de leucócitos humanos que mediam ADCC incluem células mononucleares sanguíneas periféricas (PBMC), células matadoras naturais (NK), monócitos, células T citotóxicas e neutrófilos; de modo que PBMCs e células NK são preferidas. As células efetoras podem ser isoladas a partir de uma fonte natural, por exemplo, a partir do sangue.
[244] Os termos “receptor de Fc” ou “FcR” descrevem um receptor que se liga a uma região Fc de um anticorpo. O FcR preferido é um FcR de sequência nativa humana. Além disso, um FcR preferido é aquele que se liga a um anticorpo IgG (um receptor gama) e inclui receptores das subclasses FcyRI, FcyRII e FcyRIII, incluindo variantes alélicas e, alternativamente, formas divididas desses receptores. Receptores FcyRII incluem FcyRIIA (um "receptor de ativação") e FcyRIIB (um "receptor de inibição"), que possuem sequências similares de aminoácidos que diferem entre si principalmente em seus domínios citoplasmáticos. O receptor de ativação FcyRIIA possui um motivo de ativação com base em imunoreceptor tirosina (ITAM), no seu domínio citoplasmático. O receptor de inibição FCYRIIB possuí um motivo de inibição com base em imunoreceptor tirosina (ITIM) no seu domínio citoplasmático. (vide revisão em M. Daêron, Annu. Rev. Immunol. 15:203-234 (1997)). FcRs são revisados em Ravetch e Kinet, Annu. Rev. Immunol 9:457-92 (1991); Capel et al., Immunomethods 4:25-34 (1994); e deHaas et al.et al..., J. Lab. Clin. Med. 126:330-41 (1995). Outras FCRs, incluindo aquelas a serem identificadas no futuro, são englobadas pelo termo “FcR” na presente invenção. O termo também inclui o receptor neonatal, FcRn, que é responsável pela transferência de IgGs maternos ao feto (Vide Guyer et al., J. Immunol. 117:587 (1976) e Kim et al., J. Immunol. 24:249(1994)), e regula a homeostase das imunoglobulinas. A publicação WO 2000/42072 (Presta) descreve variantes de anticorpos com ligação aos FcRs melhorada ou diminuída. O conteúdo de tais publicações de patentes é especificamente incorporado ao presente pela referência. Vide também, por exemplo, Shields et al. J. Biol. Chem. 9(2):6591-6604 (2001).
[245] Os métodos para medição da ligação ao FcRn são conhecidos (vide, por exemplo, Ghetie 1997, Hinton, 2004). A ligação ao FcRn humano in vivo e a meia vida no soro dos polipeptídeos que se liga à FcRn humano com alta afinidade de ligação podem ser analisadas, por exemplo, em camundongos transgênicos ou transfectados com linhagens de células humanas expressando o FcRn humano, ou administrado em primatas com polipeptídeos variantes de Fc.
[246] “Citotoxicidade dependente de complemento” ou “CDC” refere-se a lise de uma célula alva na presença de complemento. A ativação da via clássica do sistema complemento é iniciada pela ligação do primeiro componente do sistema de complemento (C1q) ao anticorpo (de subclasse apropriada) que está ligada ao seu antígeno cognato. Para avaliar a ativação do complemento, pode ser realizado um ensaio de CDC, conforme descrito, por exemplo, em Gazzano-Santoro et al., J. Immunol. Methods 202:163 (1996).
[247] Variantes de polipeptídeos com sequências de aminoácidos da região Fc alteradas e o aumento ou diminuição da capacidade de ligação ao C1q são descritos na Patente US 6.194.551 B1, e publicação WO 99/51642. O conteúdo dessas publicações de patentes é especificamente incorporado ao presente pela referência. Vide, também, Idusogie et al. J. Immunol. 164: 41784184 (2000).
[248] A expressão "polipeptídeo compreendendo região Fc” refere-se a um polipeptídeo, tal como um anticorpo ou imunoadesina, que compreende uma região Fc. A lisina C-terminal (resíduo 477 de acordo com sistema de numeração EU) da região Fc pode ser removida, por exemplo, durante a purificação do polipeptídeo, ou pela construção de um ácido nucleico recombinante que codifica o polipeptídeo. Consequentemente, uma composição compreendendo um polipeptídeo contendo uma região Fc de acordo com a presente invenção pode compreender polipeptídeos com K447, com todos os resíduos K447 removidos, ou uma mistura de anticorpos com e sem resíduos K447.
[249] Uma "região estrutural humana aceptora" para os propósitos da presente invenção é uma região estrutural contendo a sequência de aminoácidos de uma região estrutural VL ou VH derivada de uma região estrutural da imunoglobulina humana, ou a partir de uma região estrutural de consenso humano. Uma região estrutural humana aceptora "derivada de" uma região estrutural de imunoglobulina humana ou região estrutural de consenso humano pode compreender a mesma sequência de aminoácido desta ou pode conter uma alteração na sequência de aminoácidos pré-existente. Quando alterações de aminoácidos pré-existentes estão presentes, preferivelmente não mais de 5 e preferivelmente 4 ou menos, ou 3 ou menos alterações de aminoácidos pré-existentes estão presentes. Quando as alterações de aminoácidos pré-existentes estão presentes em um VH, essas alterações estão preferencialmente em apenas três, duas, ou uma das posições 71H, 73H e 78h, por exemplo, os resíduos de aminoácidos nessas posições podem ser 71A, 73T e/ou 78A. Em um exemplo de realização, a região estrutural humana aceptora VL é idêntica em sua sequência com a sequência da região estrutural da imunoglobulina humana VL ou sequência da região estrutural de consenso humano.
[250] Uma "região estrutural de consenso humano" é uma região estrutural que representa os mais comumente resíduos de aminoácidos que ocorrem em uma seleção de sequências da região estrutural VH ou VL da imunoglobulina humana. De modo geral, a seleção das sequências VH ou VL da imunoglobulina humana ocorre a partir de um subgrupo de sequências de domínios variáveis. De modo geral, o subgrupo de sequências é um subgrupo como em Kabat et al. Em um exemplo de realização, para o VL, o subgrupo é subgrupo kappa I como em Kabat et al. Em um exemplo de realização, para o VH, o subgrupo é subgrupo III como em Kabat et al.
[251] Uma “região estrutural de consenso VH subgrupo III” compreende a sequência de consenso obtida a partir das sequências de aminoácidos no subgrupo III variável pesado de Kabat et al. supra. Em um exemplo de realização, a sequência de aminoácidos da região estrutural de consenso VH subgrupo III compreende pelo menos uma parte ou a totalidade de cada uma das seguintes sequências: EVQLVESGGGLVQPGGSLRLSCAAS (SEQ ID NO:184)-H1-WVRQAPGKGLEWV (SEQ ID NO:185)-H2-RFTISRDNSKNTLYLQMNSLRAEDTAVYYC (SEQ ID NO:186)-H3-WGQGTLVTVSS (SEQ ID NO:187).
[252] Uma “região estrutural de consenso VL subgrupo I” compreende a sequência de consenso obtida a partir das sequências de aminoácidos no subgrupo I kappa variável leve de Kabat et al. supra. Em um exemplo de realização, a sequência de aminoácidos da região estrutural de consenso VH subgrupo I compreende pelo menos uma parte ou a totalidade de cada uma das seguintes sequências:DIQMTQSPSSLSASVGDRVTITC (SEQ ID NO:188)-L1-WYQQKPGKAPKLLIY (SEQ ID NO:189)-L2-GVPSRFSGSGSGTDFTLTISSLQPEDFATYYC (SEQ ID NO:190)-L3-FGQGTKVEIK (SEQ ID NO:191).
[253] Conforme utilizado na presente invenção, as expressões “anticorpo mutante” ou “variante de anticorpo” referem-se a uma sequência de aminoácidos variante de um anticorpo no qual um ou mais resíduos de aminoácidos do anticorpo espécie-dependente foram modificados. Tais mutantes necessariamente possuem menos de 100% identidade de sequência ou similaridade com o anticorpo espécie-dependente. Em um exemplo de realização, o anticorpo mutante possuirá uma sequência de aminoácidos que contém pelo menos 75% de identidade ou similaridade na sequência de aminoácidos com a sequência de aminoácidos de um domínio variável de cadeia leve ou pesada do anticorpo espécie-dependente, mais preferencialmente pelo menos 80%, mais preferencialmente pelo menos 85%, mais preferencialmente pelo menos 90%, e mais preferencialmente ainda pelo menos 95%. A identidade ou similaridade com relação a esta sequência é definida no presente como a percentagem de resíduos de aminoácidos na sequência candidata que é idêntica (ou seja, o mesmo resíduo) ou similar (ou seja, resíduos de aminoácido do mesmo grupo com base nas propriedades da cadeia lateral, vide abaixo) com os resíduos do anticorpo espécie-dependente, após o alinhamento das sequências e a introdução das lacunas, se necessário, para atingir a percentagem máxima de identidade de sequência. Nenhuma deleção ou inserção nas extremidades N- terminal, C-terminal ou em extensões internas da sequência de anticorpos fora do domínio variável deve ser interpretada como alteração que afeta a identidade ou similaridade da sequência.
[254] Um "distúrbio" ou “doença” é qualquer condição que possa se beneficiar de um tratamento com uma substância/molécula ou método da presente invenção. Isto inclui distúrbios ou doenças agudas e crônicas incluindo aquelas condições patológicas que predispõem o mamífero ao distúrbio em questão. Exemplos não limitantes de distúrbios que podem ser tratados na presente invenção, incluem tumores malignos e benignos; carcinoma, blastoma e sarcoma.
[255] O termo "tratamento" refere-se tanto a tratamento terapêutico quanto a medidas profiláticas ou preventivas. Aqueles que necessitam de tratamento incluem indivíduos que já possuem um tumor benigno, estado pré-canceroso, ou tumor não metastático, bem como aqueles em que a ocorrência ou recorrência do câncer deve ser evitada.
[256] O termo "quantidade terapeuticamente eficaz" refere-se a uma quantidade de um agente terapêutico para tratar ou prevenir uma doença ou distúrbio em um mamífero. No caso de cânceres, a quantidade terapeuticamente eficaz do agente terapêutico pode reduzir o número de células cancerosas, reduzir o tamanho do tumor primário; inibir (ou seja, reduzir até certo ponto e, preferencialmente, parar) a infiltração de células do câncer em órgãos periféricos; inibir (ou seja, reduzir até certo ponto e, de preferência, parar) a metástase tumoral, inibir, em alguma extensão, o crescimento tumoral e/ou aliviar em alguma extensão, um ou mais sintomas associados ao distúrbio. À medida que a droga pode impedir o crescimento e/ou matar as células cancerígenas já existentes, ela pode ser considerada citostática e/ou citotóxica. Na terapia para o câncer, a eficácia pode in vivo pode ser mensurada, por exemplo, avaliando o tempo de progressão da doença (TTP) e/ou determinando a taxa de resposta (RR), duração de resposta e/ou qualidade de vida.
[257] Os termos "câncer" e "canceroso" referem-se, ou descrevem a condição fisiológica em mamíferos que normalmente é caracterizada pelo crescimento de células de maneira não regulada. Ficam incluídos nesta definição os cânceres benignos e malignos. Por "câncer em estágio precoce" ou "tumor em fase inicial" entende-se um câncer que não é invasivo ou metastático, ou é classificado como um câncer de estagio 0, I ou II. Exemplos de câncer incluem, mas não estão limitados a, carcinoma, linfoma, blastoma (incluindo meduloblastoma e retinoblastoma), sarcoma (incluindo lipossarcoma e sarcoma de células sinoviais), os tumores neuroendócrinos (incluindo tumores carcinóides, gastrinoma e câncer de células da ilhota), mesotelioma, schwannoma (incluindo neuroma acústico), meningioma, adenocarcinoma, melanoma e leucemia linfóide ou malignidades linfóides. Exemplos mais específicos de tais cânceres incluem o câncer de células escamosas (por exemplo, câncer de células epiteliais escamosas), câncer de pulmão incluindo o câncer de pulmão de pequenas células (SCLC) e o câncer de pulmão de não pequenas células (NSCLC), adenocarcinoma de pulmão e carcinoma escamoso de pulmão, câncer de peritônio, câncer hepatocelular, câncer gástrico ou de estômago incluindo o câncer gastrointestinal, câncer pancreático, glioblastoma, câncer de colo do útero, câncer de ovário, câncer hepático, câncer de bexiga, hepatoma, câncer de mama (incluindo câncer de mama metastático), câncer de cólon, câncer de reto, câncer colo retal, câncer de endométrio ou carcinoma uterino, carcinoma de glândulas salivares, câncer de rim ou câncer renal, câncer de próstata, cânceres vulvares, câncer da tiroide, carcinoma hepático, carcinoma anal, carcinoma de pênis, câncer testicular, câncer esofagiano, tumores das vias biliares, bem como câncer de cabeça e pescoço e mieloma múltiplo.
[258] O termo “pré-canceroso” refere-se a uma condição ou um crescimento que geralmente precede ou evolui para um câncer. Um crescimento “pré-canceroso” terá células que são caracterizadas pela regulação anormal do ciclo celular, proliferação e diferenciação, que pode ser determinado por meio de marcadores da regulação do ciclo celular, proliferação celular ou diferenciação.
[259] Por “displasia” significa qualquer crescimento anormal ou desenvolvimento de tecidos, órgãos ou células. Preferencialmente, a displasia é de alto grau ou pré-cancerosa.
[260] Por "metástase" entende-se a propagação do câncer a partir de seu local primário para outros lugares do corpo. As células cancerosas podem fugir do tumor primário, penetrar nos vasos linfáticos e sanguíneos, circular através da corrente sanguínea, e crescer em um foco distante (metástases) em tecidos normais de outras partes do corpo. A metástase pode ser local ou distante. A metástase é um processo sequencial, depende de células tumorais que partem do tumor primário, viajando através da corrente sanguínea, e parando em um local distante. No novo local, as células estabelecem um suprimento de sangue e podem crescer até formar uma massa potencialmente letal.
[261] Ambas as vias moleculares (estimulatória e inibitória) dentro da célula do tumoral regulam esse comportamento, e as interações entre a célula tumoral e as células hospedeiras no local distantes também são significativas.
[262] A expressão "não metastática" significa um câncer que é benigno ou que permanece no local primário e que não tenha penetrado no sistema linfático ou vasos sanguíneos ou que não foi para outros tecidos além do local primário. Geralmente, o câncer não metastático é qualquer tipo de câncer que está e um estágio 0, I ou II e ocasionalmente é um câncer que está em um estágio III.
[263] Por "tumor primário" ou "câncer primário" entende-se o câncer original e não uma lesão metastática localizada em outro tecido, órgão ou localização no corpo do paciente.
[264] Por "tumor benigno" ou "câncer benigno" entende-se um tumor que permanece localizado no local de origem e não tem a capacidade de infiltrar, invadir ou causar metástases para um local distante.
[265] Por "carga tumoral" entende-se o número de células de câncer, o tamanho de um tumor, ou a quantidade de câncer no corpo. A carga tumoral é também referida como a carga de tumor.
[266] "Número de tumor" quer dizer a quantidade de tumores.
[267] Por "indivíduo" entende-se um mamífero, incluindo, mas não se limitando a, um mamífero humano ou não humanos, tais como bovinos, equinos, caninos, ovinos ou felinos. Preferencialmente, o indivíduo é um ser humano.
[268] O termo "terapia anticâncer" refere-se a uma terapia útil no tratamento de câncer. Exemplos de agentes terapêuticos anticâncer incluem, mas não se limitam a, por exemplo, agentes quimioterápicos, agentes inibidores do crescimento, agentes citotóxicos, agentes usados na terapia de radiação, agentes anti-angiogênicos, agentes apoptóticos, agentes anti-tubulina, e outros agentes para o tratamento do câncer, anticorpos anti-CD20, inibidores do fator de crescimento derivado de plaquetas (por exemplo, Gleevec® (mesilato de imatinibe)), um inibidor da COX-2 (por exemplo, o celecoxib), interferons, citocinas, antagonistas (por exemplo, anticorpos neutralizantes) que se liga a um ou mais dos seguintes alvos ErbB2, ErbB3 , ErbB4, PDGFR-beta, BlyS, APRIL, BCMA ou receptor(es) VEGF, TRAIL/Apo2, e outros agentes químicos bioativos e orgânicos e etc. Combinações destes são também contempladas pela presente invenção.
[269] A expressão “agente citotóxico”, da forma utilizada na presente invenção, indica uma substância que inibe ou previne a função de células e/ou causa a destruição de células. O termo é utilizado para incluir isótopos radioativos (por exemplo, I131, I125, Y90 e Re186), agentesquimioterápicos e toxinas, tais como toxinas enzimaticamente ativa de bactérias, fungos, plantas, ou de origem animal, ou fragmentos destes.
[270] Um "agente quimioterápico" é um composto químico útil no tratamento do câncer. Exemplos de agente quimioterápico incluem compostos químicos úteis no tratamento do câncer. Exemplos de agentes quimioterápicos incluem agentes alquilantes, tais como tiotepa e ciclofosfamida CYTOXAN®; sulfonatos de alquila, tais como busulfan, improsulfan e piposulfan; aziridinas, tais como benzodopa, carboquona, meturedopa e uredopa; etilenoiminas e metilamelaminas, incluindo altretamina, trietilenomelamina,trietilenofosforamida, trietilenotiofosforamida e trimetilolomelamina;acetogeninas (especialmente bulatacin e bulatacinona); camptotecina (incluindo o análogo sintético topotecan; briostatina; calistatina); CC-1065 (incluindo seus análogos sintéticos adozelesina, carzelesina e bizelesina); criptoficinas (particularmente a criptoficina 1 e criptoficina 8); dolastatina; duocarmicina (incluindo o análogo sintético, KW-2189 e CB1-TM1); eleuterobina; pancratistatina; sarcodictiina; espongistatina; mostardas de nitrogênio, tais como clorambucil, clornafazina, colofosfamida, estramustina, ifosfamida, mecloretamina, cloridrato óxido de mecloretamina, melfalan, novembiquin, fenesterina, prednimustina, trofosfamida, mostarda de uracila; nitrossuréias, tais como carmustina, clorozotocin, fotemustina, lomustina, nimustina, ranimustina; antibióticos, tais como os antibióticos de enedina (por exemplo, caliqueamicin, especialmente caliqueamicin gama1I e caliquemicin omegaI1; vide, por exemplo, Agnew, Chem. Intl. Ed. Engl., 33: 183-186 (1994); dinemicina, incluindodinemicina A; biofosfonados, tais como clodronato, esperamicina; bem como neocarzinostatina cromóforo e cromóforos antibióticos de enedina cromoproteína relacionadas), aclacinomisinas, actinomicina, autramicina, azaserina, bleomicinas, cactinomicin, carabicina, carminomicina, carzinofilina, cromomicinas, dactinomicina, daunorrubicina, detorubicina, 6-diazo-5-oxo-L- norleucina, doxorrubicina doxorubicina (incluindo ADRIAMYCIN®, morfolino- doxorubicina, cianomorfolino-doxorubicina, 2-pirrolino-doxorubicina e desoxidoxorubicina), epirubicina, esorubicina, idarubicina, marcelomicina, mitomicinas como, por exemplo, mitomicina C, ácido micofenólico, nogalamicina, olivomicinas, peplomicina, potfiromicina, puromicina, quelamicina, rodorrubicina, estreptonigrina, estreptozocina, tubercidina, ubenimex, zinostatina, zorubicina; antimetabólitos, tais como metotrexato e 5-fluorouracil (5-FU); análogos de ácido fólico, tais como denopterin, metotrexato, pteropterin, trimetrexato; análogos de purina, tais como fludarabina, 6-mercaptopurina, tiamiprina, tioguanina; análogos de pirimidina, tais como ancitabina, azacitidina, 6-azauridina, carmofur, citarabina, dideoxiuridina, doxifluridina, enocitabina, floxuridina; andrógenos, tais como calusterona, propionato de dromostanolona, epitiostanol, mepitiostano, testolactona; anti-adrenais, tais como aminoglutetimida, mitotano, trilostano; reabastecedor de ácido fólico, tal como ácido frolínico; aceglatona; aldofosfamido glicosídeo; ácido aminolevulínico; amsacrina; bestrabucil; bisantreno; edatraxato; defofamina; demecolcina; diaziquona; elfornitina; acetato de eliptínio; epotilona; etoglucídeo; nitrato de gálio; hidroxiuréia; lentinan; lonidamina; maitansinóides, tais como maitansina e ansamitocinas; mitoguazona; mitoxantrona; mopidamol; nitacrina; pentostatina; fenamet; pirarubicina; ácido podofilínico; 2-etilidrazida; procarbazina; PSK® complexo polissacarídico (JHS Natural Products, Eugene, OR); razoxano; rizoxin; sizofiran; espirogermânio; ácido tenuazônico; triaziquona; 2,2’,2”-triclorotrietilamina; tricotecenos (especialmente toxina T-2, verracurin A, roridin A e anguidina); uretano; vindesina; dacarbazina; manomustina; mitobronitol; mitolactol; pipobroman; gacitosina; arabinosida (“Ara-C); ciclofosfamida; tiotepa; taxóides, tais como, paclitaxel TAXOL® (Bristol-Myers Squibb Oncology, Princeton, N.J.), formulação de nanopartículas de paclitaxel construidas em albumina ABRAXANE® (American Pharmaceutical Partners, Schaumberg, Illinois), e doxetaxel TAXOTERE® (Rhône-Poulenc Rorer, Antony, França); clorambucil, GEMZAR® gencitabina; 6-tioguanina; mercaptopurina; metotrexato; análogos de platina tais como cisplatina e carboplatina; vinblastina; platina; etoposida (VP- 16); ifosfamida; mitoxantrona; vincristina; NAVELBINE® vinorelbine; novantrona; teniposide, edatrexato; daunomicina; aminopterina; xeloda, ibandronato; irinotecano (Camptosar, CPT-11) (incluindo o regime de tratamento de irinotecano com 5-FU e leucovorina), inibidor de topoisomerase RFS 2000; difluorometilornitina (DMFO); ácido retinóico; capecitabina; combretastatina; VELCADE bortezomib; REVLIMID lenalidomide; leucovorin (LV); oxaliplatina, incluindo o regime de tratamento oxaliplatina (FOLFOX); inibidores da PKC-alfa, Raf, H-Ras e EGFR (por exemplo, o erlotinib (Tarceva®) e EVGF-A) que reduz a proliferação celular e os sais, ácidos ou derivados farmaceuticamente aceitáveis de quaisquer dos acima mencionados.
[271] Também estão incluídos nesta definição os agentes anti- hormonais que agem regulando ou inibindo a ação hormonal tal como antiestrógenos e os moduladores seletivos do receptor do estrógeno (SERMs), incluindo, por exemplo, tamoxifeno (incluindo o tamoxifeno NOLVADEX®, raloxifeno, droloxifeno, 4-hidroxitamoxifeno, trioxifeno, keoxifeno, LY117018, onapristona, e toremifeno FARESTON®); inibidores da aromatase que inibem enzima aromatase, que regula a produção do estrógeno nas glândulas adrenais como, por exemplo, 45-imidazoles, aminoglutetimida, acetato de megestrol MEGASE®, exemestane AROMASIN®, formestanie, fadrozole, vorozole RIVISOR®, letrozole FEMARA®, e anastrozole ARIMIDEX® e anti-andrógenos, tais como flutamida, nilutamida, bicalutamida, leuprolida e goserelina, bem como troxacitabina um nucleosídeo 1,3-dioxolano análogo a citosina; oligonucleotídeos antisense, particularmente aqueles que inibem a expressão dos genes nas vias de sinalização envolvida na proliferação celular aberrante, como, por exemplo, PKC-alfa, Raf e H-Ras; ribozimas, tal como um inibidor da expressão do VEGF (por exemplo, ribozima ANGIOZYME®) e um inibidor da expressão de HER2, vacinas como as vacinas para terapia gênica, por exemplo, vacina ALLOVECTIN®, vacina LEUVECTIN ® e vacina VAXID®; PROLEUKIN® rIL-2; LURTOTECAN®; inibidor da topoisomerase 1; ABARELIX® rmRH; Vinorelbina e Esperamicinas (vide Patente US 4.675.187), e sais farmaceuticamente aceitáveis, ácidos ou derivados de qualquer um dos compostos acima.
[272] O termo “pró-droga”, conforme utilizado na presente invenção, designa uma forma precursora ou derivativa de uma substância farmaceuticamente ativa que é menos citotóxica para células tumorais em comparação com a droga parental e é capaz de ser enzimaticamente ativada ou convertida na forma parental mais ativa. Vide, por exemplo, Wilman, “Prodrugs in Cancer Chemotherapy" Biochemical Society Transactions, 14, págs. 375-382, 615a Reunião de Belfast (1986) e Stella et al., Prodrugs: A Chemical Approach to Targeted Drug Delivery, Directed Drug Delivery, Borchardt et al., (ed.), págs. 247-267, Human Press (1985). As pró-drogas de acordo com a presente invenção incluem, mas não se limitam a, pró-drogas que contêm fosfato, pró- drogas que contêm tiofosfato, pró-drogas que contêm sulfato, pró-drogas que contêm peptídeos, pró-drogas modificadas com D-aminoácidos, pró-drogas glicosiladas, pró-drogas que contêm beta-lactama, pró-drogas que contêm fenoxiacetamida opcionalmente substituída ou pró-drogas que contêm fenilacetamida opcionalmente substituída, 5-fluorocitosina e outras pró-drogas de 5-fluorouridina que podem ser convertidas na droga livre citotóxica mais ativa. Exemplos de drogas citotóxicas que podem ser derivatizadas na forma de pró- droga para uso na presente invenção incluem, mas sem limitar-se a, aqueles agentes quimioterápicos descritos anteriormente.
[273] O termo "radioterapia" significa o uso de raios gama ou raios beta direcionados para induzir um dano suficiente em uma célula, de modo a limitar a sua capacidade de funcionar normalmente ou para destruir a célula completamente. Será apreciado que existem diversas maneiras conhecidas no estado da técnica para determinar a dose e a duração do tratamento. Os tratamentos típicos são fornecidos por uma administração única e as doses típicas variam de 10 a 200 unidades (Grays) por dia.
[274] Uma “amostra biológica” (denominada de maneira intercambiável como “amostra” ou “amostra de tecido ou célula”) abrange uma variedade de tipos de amostras obtidas a partir de um indivíduo e pode ser utilizada em um teste diagnóstico ou ensaio de acompanhamento. A definição abrange sangue e outras amostras líquidas de origem biológica, amostras de tecidos sólidos, tal como biópsia ou culturas de tecidos ou células derivadas destes, e a progênie destas. A definição também inclui amostras que foram manipuladas de qualquer forma após a sua obtenção, como por tratamento com reagentes, solubilização, ou enriquecimento de certos componentes, como proteínas ou polinucleotídeos, ou emblocadas em uma matriz semi-sólida ou sólida para fins de corte. A expressão “amostra biológica” abrange uma amostra clínica, e também inclui as células em cultura, sobrenadantes de células, lisados celulares, soros, plasmas, fluidos biológicos e amostras de tecido. A fonte da amostra biológica pode ser o tecido solide como a partir de uma amostra de órgão ou tecido fresco, congelado e/ou conservados ou a partir de biópsia ou aspirado; sangue ou os componentes do sangue, fluidos corporais, tais como liquor, líquido amniótico, fluido peritoneal ou líquido intersticial; células a partir de qualquer momento da gestação ou desenvolvimento do indivíduo. Em alguns exemplos de realização, a amostra biológica é obtida a partir de um tumor primário ou metastático. A amostra biológica pode conter compostos que não estão naturalmente misturadas com o tecido na natureza, tais como conservantes, anticoagulantes, tampões, fixadores, nutrientes, antibióticos e similares.
[275] Para os propósitos da presente invenção, “corte” ou “secção” de uma amostra de tecido significa uma única porção ou um pedaço de uma amostra de tecido, por exemplo, uma fatia fina de tecido ou células cortadas a partir de uma amostra de tecido. Entende-se que várias secções de amostras de tecido podem ser obtidas e submetidas à análise de acordo com a presente invenção. Em alguns exemplos de realização, a mesma secção de amostra de tecido é analisada, tanto a nível morfológico como molecular, ou é analisada com relação a proteínas e ácidos nucleicos.
[276] A palavra "marcador", quando utilizada no presente refere- se à um composto ou composição que está conjugada ou fundida direta ou indiretamente a um reagente como uma sonda de ácido nucleico ou um anticorpo e facilita a detecção do reagente ao qual está conjugado ou fundido. O marcador pode ser ele mesmo detectável (por exemplo, marcadores radioisótopos ou marcadores fluorescentes), ou, no caso de um marcador enzimático, pode catalisar uma alteração química de um composto ou composição de substrato que é detectável.
[277] A presente invenção abrange composições, incluindo composições farmacêuticas, compreendendo um anticorpo anti-FGFR3, e polinucleotídeos compreendendo sequências que codificam um anticorpo anti- FGFR3. Conforme utilizado no presente, composições compreendem um ou mais anticorpos que se ligam a FGFR3, e/ou um ou mais polinucleotídeos compreendendo sequências de codificação de um ou mais anticorpos que se ligam a FGFR3. Estas composições podem ainda compreender veículos adequados, tais como excipientes farmaceuticamente aceitáveis, incluindo tampões, que são bem conhecidos no estado da técnica.
[278] A presente invenção também abrange realizações de anticorpos e polinucleotídeos isolados. A presente invenção também abrange realizações de anticorpos e polinucleotídeos substancialmente puros.
[279] A presente invenção também engloba um método para tratar uma doença, por exemplo, mieloma múltiplo ou carcinoma na fase de transição (por exemplo, carcinoma invasivo na fase de transição) com um anticorpo anti- FGFR3 (conforme descrito no presente ou conforme é conhecido no estado da técnica).
[280] Os anticorpos anti-FGFR3 da presente invenção são preferencialmente monoclonais. Estão também dentro do escopo da presente invenção fragmentos Fab, Fab', Fab'-SH e F(ab')2 dos anticorpos anti-FGFR3 fornecidos na presente invenção. Estes fragmentos de anticorpos podem ser gerados por meios tradicionais, tal como por digestão enzimática, ou podem ser gerados por técnicas recombinantes. Tais fragmentos de anticorpos podem ser quiméricos ou humanizados. Esses fragmentos são úteis para os fins diagnósticos e terapêuticos definidos abaixo.
[281] Os anticorpos monoclonais são obtidos a partir de uma população de anticorpos substancialmente homogênea, ou seja, os anticorpos individuais compreendendo a população são idênticos, exceto quanto a possíveis mutações de ocorrência natural que podem estar presentes em pequenas quantidades. Assim, o adjetivo “monoclonal” indica o caráter do anticorpo como não sendo obtido a partir de uma mistura de anticorpos distintos.
[282] Os anticorpos monoclonais anti-FGFR3 da presente invenção podem ser fabricados utilizando o método de hibridoma descrito primeiramente por Kohler et al., Nature 256: 495 (1975) ou podem ser produzidos por meio de métodos de DNA recombinante (Patente US 4.816.567).
[283] No método de hibridoma, um camundongo ou outro animal hospedeiro adequado, tal como o hamster, é imunizado para suscitar os linfócitos que produzem ou são capazes de produzir anticorpos que se ligam de forma especifica a proteína utilizada para imunização. Os anticorpos para FGFR3 são elevados nos animais por meio de múltiplas injeções subcutâneas (sc) ou intraperitoneais (ip) de FGFR3 e um adjuvante. O FGFR3 pode ser preparado usando métodos conhecidos no estado da técnica, alguns dos quais estão descritos no presente documento. Por exemplo, a produção recombinante de FGFR3 humano e de camundongo está descrita abaixo. Em um exemplo de realização, os animais são imunizados com um FGFR3 fundido a uma porção Fc de uma cadeia pesada de imunoglobulina. Em um exemplo de realização preferido, os animais são imunizados com uma proteína de fusão FGFR3-IgG1. Os animais normalmente são imunizados contra conjugados imunogênicos ou derivados de FGFR3 com monofosforil lipídio A (MPL)/dicrinomicolato de trealose (TDM) (Ribi Immunochem. Inc., Hamilton, MT) e a solução é injetada por via intradérmica em vários locais. Duas semanas depois, os animais são submetidos a imunizações intensificadoras (boosted). De 7 a 14 dias mais tarde os animais são sangrados e o soro é testado quanto ao título de anticorpo anti- FGFR3. Os animais são submetidos a imunização intensificadora até atingir o título platô.
[284] Alternativamente, linfócitos podem ser imunizados in vitro. Os linfócitos são fundidos em seguida com células de mieloma, utilizando um agente de fusão apropriado, tal como o polietilenoglicol, para formar uma célula de hibridoma (Goding, Monoclonal Antibodies: Principles and Practice, págs. 59 a 103 (Academic Press, 1986)).
[285] As células de hibridoma assim preparadas podem ser semeadas e cultivadas em um meio de cultura apropriado que contém uma ou mais substâncias que inibem o crescimento ou a sobrevivência das células de mieloma parentais não fundidas. Por exemplo, se as células de mieloma parentais não possuem a enzima hipoxantina-guanina-fosforribosiltransferase (HGPRT ou HPRT), o meio de cultura para os hibridomas incluirá tipicamente hipoxantina, aminopterina e timidina (meio HAT), substâncias que evitam o crescimento de células com deficiência em HGPRT.
[286] Células de mieloma preferidas são aquelas que se fundem eficientemente, suportam a produção estável em alto nível de anticorpos pelas células produtoras de anticorpos selecionadas e são sensíveis a um meio, tal como meio HAT. Entre estas, as linhagens de células de mieloma mais preferidas são linhagens de mieloma murino, tais como as derivadas de tumores de camundongos MOPC-21 e MPC-11 que podem ser obtidas no Salk Institute Cell Distribution Center, San Diego, Califórnia, Estados Unidos, células SP-2 ou X63-Ag8-653 disponíveis pelo American Type Culture Collection, Rockville, Maryland USA. Linhagens celulares de mieloma humano e heteromieloma humano/camundongo também foram descritas para a produção de anticorpos monoclonais humanos (Kozbor, J. Immunol., 133:3001 (1984); e Brodeur et al., Monoclonal Antibody Production Techniques and Applications, págs. 51-63 (Marcel Dekker, Inc., Nova Iorque, 1987)).
[287] O meio de cultura em que são cultivadas células de hibridoma é testado para determinar a produção de anticorpos monoclonais direcionados contra o FGFR3-. Preferencialmente, a especificidade de ligação de anticorpos monoclonais produzidos pelas células de hibridoma é determinada pela imunoprecipitação ou por um ensaio de ligação in vitro, tais como o radioimunoensaio (RIA) ou ensaio imunoenzimático (ELISA).
[288] A afinidade de ligação do anticorpo monoclonal pode ser determinada, por exemplo, através da análise de Scatchard de Munson et al., Anal. Biochem., 107: 220 (1980).
[289] Após a identificação de células de hibridoma que produzem anticorpos de especificidade, afinidade e/ou atividade desejadas, os clones podem ser subclonados por limitação com procedimentos de diluição e cultivados por meio de métodos padrões (Goding, Monoclonal Antibodies: Principles and Practice, págs.59-103 (Academic Press, 1986)). Meios de cultura apropriados para este propósito incluem, por exemplo, meio D-MEM ou RPMI- 1640. Além disso, as células de hibridoma podem ser cultivadas in vivo na forma de tumores ascíticos em um animal.
[290] Os anticorpos monoclonais secretados pelos subclones são apropriadamente separados do meio de cultura, liquido ascítico ou soro por meio de procedimentos convencionais de purificação de imunoglobulina, tais como, por exemplo, proteína A-Sefarose, cromatografia de hidroxilapatita, eletroforese em gel, diálise ou cromatografia por afinidade.
[291] Os anticorpos anti-FGFR3 da presente invenção podem ser produzidos utilizando bibliotecas combinatórias para a procura de clones de anticorpos sintéticos com a(s) atividade(s) desejada(s). Em princípio, os clones sintéticos de anticorpos são selecionados pela triagem de bibliotecas de fago contendo fagos que exibem vários fragmentos da região variável de anticorpos (Fv) fundidos na proteína da cápside do fago. Essas bibliotecas de fago são testadas por cromatografia de afinidade contra o antígeno desejado. Clones expressando fragmentos Fv capazes de se ligar ao antígeno desejado são absorvidos pelo antígeno e, assim, separados dos clones que não se ligam na biblioteca. Os clones que se ligam são então eluidos do antígeno e podem ainda ser enriquecidos por ciclos adicionais de adsorção/eluição de antígenos. Qualquer um dos anticorpos anti-FGFR3 da presente invenção pode ser obtido pela concepção de um procedimento de seleção adequado para selecionar o clone fago de interesse seguido pela construção de um anticorpo anti-FGFR3 de comprimento total usando as sequências Fv a partir do clone de fago de interesse e a sequência adequada da região constante (FC) descrita em Kabat et al., Sequences of Proteins of Immunological Interest, quinta edição, NIH Publication 91-3242, Bethesda MD (1991), vols. 1-3.
[292] O domínio de ligação ao antígeno de um anticorpo é formado por duas regiões variáveis (V) de cerca de 110 aminoácidos, selecionados, cada um, de cadeias leve (VL) e pesada (VH), em que ambos apresentam três alças hipervariáveis ou regiões determinantes de complementaridade (CDRs). Os domínios variáveis podem ser exibidos funcionalmente no fago, tanto como fragmentos Fv de cadeia simples (scFv), em que VH e VL são covalentemente ligados por meio de um peptídeo flexível e curto, quanto como fragmentos Fab, em que é, cada um, fundidos a um domínio constante, e interagem de forma não covalente, conforme descrito em Winter et al., Ann. Rev. Immunol., 12: 433-455 (1994). Conforme utilizado na presente invenção, os clones fagos que codificam scFv e clones fagos que codificam Fab são coletivamente referenciados como “clones de fagos Fv” ou “clones Fv”.
[293] Os repertórios de genes VH e VL podem ser clonados separadamente por meio de reação em cadeia da polimerase (PCR) e recombinados de forma aleatória em bibliotecas de fagos, que podem então ser pesquisados para os clones de ligação ao antígeno, conforme descrito em Winter et al., Ann. Rev. Immunol., 12: 433-455 (1994). Bibliotecas de fontes imunizadas fornecem anticorpos de alta afinidade para o imunógeno, sem a necessidade de construção de hibridomas. Alternativamente, o repertório inicial (naive) pode ser clonado para fornecer uma única fonte de anticorpos humanos, para uma faixa ampla de antígenos não-próprios (non-self) e também próprios (self), sem qualquer imunização, conforme descrito por Griffiths et al., EMBO J, 12: 725-734 (1993) Finalmente, bibliotecas iniciais (naive) podem também ser produzidas sinteticamente por meio de clonagem de segmentos de gene V não rearranjados a partir de células tronco, e utilizando primers de PCR que contém sequência aleatória para codificar as regiões CDR3 altamente variáveis e para realizar o rearranjo in vitro, conforme descrito por Hoogenboom e Winter, J. Mol. Biol., 227: 381-388 (1992).
[294] Fagos filamentosos são utilizados para exibir fragmentos de anticorpo pela fusão com a proteína de revestimento menor pIII. Os fragmentos de anticorpos podem ser exibidos como fragmentos Fv de cadeia simples, em que os domínios VH e VL são conectados na mesma cadeia polipeptídica por um espaçador de polipeptídeo flexível, tal como descrito por Marks et al., J. Mol. Biol., 222: 581-597 (1991), ou como fragmentos Fab, em que uma cadeia é fundida a pIII e a outra é secretada no periplasma de células hospedeiras bacterianas, onde ocorre a montagem de uma estrutura proteína de revestimento-Fab que passa a ser exibida na superfície do fago pelo deslocamento de algumas das proteínas tipo selvagem do revestimento, conforme descrito em Hoogenboom et al., Nucl. Acids Res., 19: 4133-4137 (1991).
[295] Em geral, ácidos nucleicos que codificam fragmento gênico do anticorpo são obtidos a partir de células imunes coletadas de humanos e animais. Se uma biblioteca influenciada a favor de clones anti-FGFR3 é desejada, o indivíduo é imunizado com FGFR3 para gerar uma resposta de anticorpo, e células do baço e/ou células B circundante ou outros linfócitos do sangue periférico (PBLs) são recuperados a partir da construção da biblioteca. Em uma realização preferida, uma biblioteca de fragmento de gene de anticorpo humano influenciada a favor de clones anti-FGFR3 humano é obtida pela geração de uma resposta de anticorpo anti-FGFR3 em camundongos transgênicos que carregam um arranjo gênico de imunoglobulina humana funcional (e carecem de um sistema de produção de anticorpo endógeno funcional), de modo que a imunização com FGFR3 suscita as células B a produzirem anticorpos humanos contra FGFR3. A geração de camundongos transgênicos que produzem anticorpos humanos está descrita abaixo.
[296] O enriquecimento adicional para populações de células reativas anti-FGFR3 pode ser obtida pelo uso de um procedimento de seleção apropriado, para isolar células B que expressam anticorpo de ligação à FGFR3 específico na membrana, tal como pela separação de células com afinidade de FGFR3 por cromatografia ou adsorção de células ao FGFR3 marcado com fluorocromo seguida pela seleção das células ativadas pela fluorescência (FACS).
[297] Alternativamente, o uso de células do baço e/ou células B ou outras PBLs de um doador não imunizado fornece uma melhor representação do possível repertório de anticorpo, e também permite a construção de uma biblioteca de anticorpos pelo uso de qualquer espécie animal (humana ou não humana) no qual FGFR3 não é antigênico. Para bibliotecas que incorporam construção gênica de anticorpo in vitro, as células-tronco são coletadas do indivíduo para fornecer ácidos nucleicos que codificam segmentos gênicos de anticorpo não rearranjados. As células imunes de interesse podem ser obtidas de uma série de espécies animais, tal como humano, camundongo, rato, lagomorfos, luprinos, caninos, felinos, porcos, bovinos, equinos e espécies de aves etc.
[298] Ácidos nucleicos que codificam segmentos gênicos variáveis de anticorpos (incluindo segmentos VH e VL) são recuperados de células de interesse e amplificados. No caso de bibliotecas gênicas VH e VL rearranjadas, o DNA desejado pode ser obtido pelo isolamento de DNA genômico ou mRNA de linfócitos, seguido pela reação em cadeia da polimerase (PCR) com primers que reconhecem as extremidades 5' e 3' de genes VH e VL rearranjados, conforme descrito em Orlandi et al., Proc. Natl. Acad. Sci. (USA), 86: 3833-3837 (1989), produzindo, desta forma, repertories diversos de gene V para expressão. Os genes V podem ser amplificados de cDNA e DNA genômico, com primers reversos na extremidade 5' do éxon que codifica o domínio V maduro, e primers diretos com base no segmento J, conforme descrito em Orlandi et al. (1989) e em Ward et al., Nature, 341: 544-546 (1989). Entretanto, para a amplificação de cDNA, primers reversos podem também ser baseados no éxon líder, conforme descrito por Jones et al., Biotechnol., 9: 88-89 (1991), e primers diretos na região constante, conforme descrito em Sastry et al., Proc. Natl. Acad. Sci. (USA), 86: 5728-5732 (1989). Para maximizar a complementaridade, degeneração pode ser incorporada nos primers, conforme descrito em Orlandi et al. (1989) ou Sastry et al. (1989). Preferivelmente, a diversidade da biblioteca é maximizada pelo uso de primers de PCR alvos para cada família de gene V, para amplificar todos os arranjos VH e VL disponíveis presentes na amostra de ácido nucleico de células imunes, por exemplo, conforme descrito no método de Marks et al., J. Mol. Biol., 222: 581-597 (1991), ou conforme descrito no método de Orum et al., Nucleic Acids Res., 21: 4491-4498 (1993). Para a clonagem do DNA amplificado em vetores de expressão, sítios de restrição raros podem ser introduzidos no primer de PCR como uma marca em uma extremidade, conforme descrito em Orlandi et al. (1989), ou pela amplificação por PCR adicional com um primer marcado, conforme descrito em Clackson et al., Nature, 352: 624-628 (1991).
[299] Repertórios de genes V rearranjados sinteticamente podem ser derivados in vitro de segmentos de gene V. A maior parte dos segmentos de gene VH humanos pode ser clonada e sequenciada (descrito em Tomlinson et al., J. Mol. Biol., 227: 776-798 (1992)), e mapeada (descrito em Matsuda et al., Nature Genet., 3: 88-94 (1993)); estes segmentos clonados (incluindo todas as conformações principais da alça H1 e H2) podem ser utilizados para gerar diversos repertórios de gene VH com primers de PCR que codificam alças H3 de diversas sequências e comprimentos, conforme descrito em Hoogenboom e Winter, J. Mol. Biol., 227: 381-388 (1992). Repertórios de VH podem também ser produzidos com toda a diversidade de sequência, focada em uma alça (loop) H3 longa de um único comprimento, conforme descrito em Barbas et al., Proc. Natl. Acad. Sci. USA, 89: 4457-4461 (1992). Segmentos VK e VÀ humanos foram clonados e sequenciados (descrito em Williams e Winter, Eur. J. Immunol., 23: 1456-1461 (1993)) e podem ser utilizados para produzir repertórios de cadeia leve sintéticos. Os repertórios de gene V sintéticos, com base na faixa de vezes de VH e VL, e comprimento de L3 e H3, codificarão anticorpos de considerável diversidade estrutural. Seguindo a amplificação dos DNAs que codificam o gene V, segmentos de gene V de linhagem germinativa podem ser rearranjados in vitro de acordo com métodos de Hoogenboom e Winter, J. Mol. Biol., 227: 381388 (1992).
[300] Repertórios de fragmentos de anticorpo podem ser construídos pela combinação de repertórios dos genes VH e VL juntos em diversas formas. Cada repertório pode ser criado em diferentes vetores, e os vetores recombinados in vitro, por exemplo, conforme descrito em Hogrefe et al., Gene, 128: 119-126 (1993), ou in vivo por infecção combinatória, por exemplo, o sistema loxP descrito em Waterhouse et al., Nucl. Acids Res., 21: 2265-2266 (1993). O método de recombinação in vivo explora a natureza de cadeia dupla de fragmentos Fab para superar o limite no tamanho de biblioteca imposto pela eficiência de transformação da E. coli. Repertórios VH e VL iniciais (naive) são clonados de forma separada, um em um fagomídeo e o outro em um vetor de fago. As duas bibliotecas são então combinadas por infecção de fago de bactéria contendo fagomídeo, de forma que cada célula contém uma combinação diferente, e o tamanho da biblioteca é limitado apenas pelo número de células presente (cerca de1012 clones). Ambos os vetores contêm sinais de recombinação in vivo, de forma que os genes VH e VL sejam recombinados em um único replicon, e são co-embalados em vírion de fago. Estas diversas bibliotecas fornecem um grande número de anticorpos diversos de boa afinidade (Kd-1 de cerca de 10-8 M).
[301] Alternativamente, os repertórios podem ser clonados de forma sequencial no mesmo vetor, tal como descrito em Barbas et al., Proc. Natl. Acad. Sci. USA, 88: 7978-7982 (1991), ou unidos por PCR e então clonados, conforme descrito em Clackson et al., Nature, 352: 624-628 (1991). A junção por PCR pode também ser utilizada para unir DNAs VH e VL com DNA que codifica um espaçador peptídico flexível para formar repertórios de Fv de cadeia simples (scFv). Ainda em outra técnica, a “montagem por PCR em célula” (in cell PCR assembly) é utilizada para combinar genes VH e VL nos linfócitos por PCR e então os repertórios dos genes ligados são clonados, conforme descrito em Embleton et al., Nucl. Acids Res., 20: 3831-3837 (1992).
[302] Os anticorpos produzidos por bibliotecas iniciais (naive) (tanto naturais quanto sintéticos) podem ser de afinidade moderada (Kd-1 de cerca de 106 a 107 M-1), mas maturação da afinidade pode também ser mimetizada in vitro pela construção e nova seleção de bibliotecas secundárias, conforme descrito em Winter et al. (1994), supra. Por exemplo, mutações podem ser introduzidas de forma aleatória in vitro pelo uso da polimerase passível de erro (descrito em Leung et al., Technique, 1: 11-15 (1989)) no método de Hawkins et al., J. Mol. Biol., 226: 889-896 (1992) ou no método de Gram et al., Proc. Natl. Acad. Sci USA, 89: 3576-3580 (1992). Adicionalmente, a maturação da afinidade pode ser realizada por mutação aleatória de uma ou mais CDRs, por exemplo, pelo uso de PCR com primers que carregam sequências aleatórias transportando o CDR de interesse, nos clones Fv individuais selecionados e selecionando os clones de afinidades mais altas. A publicação WO 96/07754 (publicado em 14 de março de 1996) descreveu um método para indução da mutagênese em uma região determinante de complementaridade de uma cadeia leve de imunoglobulina para criar uma biblioteca de genes de cadeia leve. Outra realização eficaz é recombinar os domínios VH ou VL selecionados pela exibição por fago com repertórios de variantes de domínio V de ocorrência natural, obtidos a partir de doadores não-imunizados, e seleção para alta afinidade em diversos ciclos de reorganização da cadeia, conforme descrito em Marks et al., Biotechnol., 10: 779-783 (1992). Esta técnica permite a produção de anticorpos e fragmentos de anticorpos com afinidades na faixa de 10-9 M.
[303] As sequências de aminoácidos e ácidos nucleicos do FGFR3 são conhecidas no estado da técnica. A sequência de ácido nucleico que codifica o FGFR3 pode ser projetada usando a sequência de aminoácidos da região do FGFR3 desejada. Como é bem conhecido no estado da técnica, há duas principais isoformas com splicing diferentes de FGFR3; o FGFR3-IIIb e FGFR3-IIIc. As sequências FGFR3 são bem conhecidas no estado da técnica e podem incluir a sequência do número de acesso da UniProKB/Swiss-Prot: P22607 (FGFR3 IIIc) ou P22607_2 (FGFR3 IIIb). As mutações de FGFR3 foram identificadas e são bem conhecidas no estado da técnica e incluem as seguintes mutações com referência às sequências exibidas na UniProKB/Swiss-Prot número de acesso P22607 (FGFR3 IIIc) ou P22607_2 (FGFR3 IIIb).
[304] Os ácidos nucleicos que codificam o FGFR3 podem ser preparados por uma variedade de métodos conhecidos no estado da técnica. Estes métodos incluem, mas não estão limitados a: síntese química por qualquer um dos métodos descritos em Engels et al. Agnew. Chem. Int. Ed. Engl, 28: 716734 (1989), tal como os métodos de triéster, fosfito, fosforamidita e H-fosfonato. Em um exemplo de realização, os códons preferidos pela célula hospedeira de expressão são utilizados no desenvolvimento do DNA que codifica FGFR3. Alternativamente, o DNA codificante de FGFR3 pode ser isolado de uma biblioteca genômica ou cDNA.
[305] Após a construção da molécula de DNA que codifica o FGFR3, a molécula de DNA é operacionalmente ligada a uma sequência de controle de expressão em um vetor de expressão, tal como um plasmídeo, de modo que a sequência de controle é reconhecida pela célula hospedeira transformada com o vetor. De modo geral, os vetores plasmídeos possuem sequências de replicação e controle que são derivadas de espécies compatíveis com a célula hospedeira. Normalmente o vetor carrega um sítio de replicação, bem como sequências que codificam proteínas que são capazes de fornecer a seleção fenotípica nas células transformadas. Os vetores adequados para a expressão nas células hospedeiras procariotas e eucariotas são conhecidos no estado da técnica e alguns estão descritos em maiores detalhes na presente invenção. Podem ser utilizados organismos eucarióticos, tal como leveduras, ou células derivadas de organismos multicelulares, tal como os mamíferos.
[306] Opcionalmente, o DNA que codifica o FGFR3 está operacionalmente ligado a uma sequência “líder de secreção” resultando na secreção do produto de expressão a partir da célula hospedeira para o meio de cultura. Exemplos de sequências líder de secreção incluem stll, ecotin, lamB, herpes GD, lpp, fosfatase alcalina, invertase e fator alfa. É também adequado para utilização na presente invenção a sequência líder aminoácido 36 da proteína A (Abrahmsen et al, EMBO J., 4: 3901 (1985)).
[307] As células hospedeiras são transfectadas e preferivelmente transformadas com os vetores de expressão ou de clonagem descritos acima na presente invenção e cultivadas em meio nutriente convencional modificado de forma a ser apropriado para a indução dos promotores, seleção de transformantes ou amplificação dos genes que codificam as sequências desejadas.
[308] Transfecção refere-se à incorporação de um vetor de expressão por uma célula hospedeira, independentemente se as sequências deste vetor são ou não de fato expressas. Diversos métodos de transfecção são conhecidos pelos técnicos hábeis no assunto, por exemplo, a precipitação em CaPO4 e eletroporação. Uma transfecção realizada com sucesso é geralmente reconhecida quando qualquer indicação da operação do vetor transfectado ocorre dentro da célula hospedeira. Os métodos de transfecção são bem conhecidos no estado da técnica, e alguns são adicionalmente descritos na presente invenção.
[309] Transformação significa a introdução de DNA em um organismo de forma que o DNA seja replicável, quer seja como um elemento extracromossômicos ou como um integrante cromossômico. Dependendo da célula hospedeira utilizada, a transformação é realizada utilizando técnicas padrões apropriadas para tais células. Os métodos de transformação são bem conhecidos no estado da técnica, e alguns são adicionalmente descritos na presente invenção.
[310] As células hospedeiras procarióticas usadas para produzir FGFR3 podem ser cultivadas conforme descrito de modo geral em Sambrook et al. Supra.
[311] As células hospedeiras de mamíferos usadas para produzir o FGFR3 podem ser cultivadas em uma variedade de meios, que são bem conhecidos no estado da técnica e alguns dos quais estão descritos na presente invenção.
[312] As células hospedeiras referidas na presente invenção abrangem células em cultura in vitro, bem como células que estão dentro de um animal hospedeiro.
[313] A purificação do FGFR3 pode ser realizada utilizando métodos reconhecidos no estado da técnica, alguns dos quais estão descritos na presente invenção.
[314] O FGFR3 purificado pode ser anexado a uma matriz adequada, tais como esferas de agarose, esferas de acrilamida, esferas de vidro, celulose, vários copolímeros acrílicos, gel de metacrilato de hidroxila, copolímeros poliacrílico e polimetacrílico, nylon, veículos neutros e iônicos, e similares para uso na separação cromatográfica por afinidade de clones exibidos em fagos. A fixação da proteína FGFR3 à matriz pode ser realizada por métodos descritos em Methods in Enzymology, vol. 44 (1976). Uma técnica comumente empregada para o acoplamento de proteínas ligantes em matrizes de polissacarídeos, por exemplo, agarose, dextrano ou celulose, envolve a ativação do transportador com haletos de cianogênio e o subsequente acoplamento de aminas aromáticas ou alifáticas primárias do peptídeo ligante à matriz ativada.
[315] Alternativamente, o FGFR3 pode ser utilizado para revestir as paredes de placas de adsorção, expresso em células hospedeiras fixadas nas placas de adsorção ou utilizado na seleção de células, ou conjugado com biotina para captura com esferas revestidas com estreptoavidina, ou utilizado em qualquer outro método conhecido no estado da técnica para selecionar bibliotecas de exibição por fago.
[316] As amostras de biblioteca de fago são colocadas em contato com o FGFR3 imobilizado sob condições apropriadas para a ligação de pelo menos uma porção das partículas de fago com o adsorvente. Normalmente, as condições, incluindo pH, força iônica, temperatura e similares, são selecionadas de forma a imitar condições fisiológicas. Os fagos ligados à fase sólida são lavados e então eluidos por ácido, tal como descrito em Barbas et al., Proc. Natl. Acad. Sci USA, 88: 7978-7982 (1991), ou por álcali, conforme descrito em Marks et al., J. Mol. Biol., 222: 581-597 (1991), ou por competição do antígeno FGFR3, por exemplo, em um procedimento similar ao método de competição de antígeno de Clackson et al., Nature, 352: 624-628 (1991). Fagos podem ser enriquecidos de 20-1.000 vezes em um único ciclo de seleção. Adicionalmente, os fagos enriquecidos podem ser crescidos em culturas bacterianas e submetidos a etapas adicionais de seleção.
[317] A eficiência de seleção depende de diversos fatores, incluindo as cinéticas de dissociação durante a lavagem, e se múltiplos fragmentos de anticorpos em um único fago podem se engajar simultaneamente com o antígeno. Anticorpos com rápida dissociação cinética (e fraca afinidade de ligação) podem ser retidos pelo uso de lavagens curtas, exibição por fago multivalente e alta densidade de revestimento de antígeno na fase sólida. A alta densidade não apenas estabiliza o fago por meio de interações multivalente, mas favorece a nova ligação do fago que foi dissociado. A seleção de anticorpos com cinéticas de dissociação lentas (e boa afinidade de ligação) pode ser promovida pelo uso de longas lavagens e exibição por fago monovalente, conforme descrito em Bass et al., Proteins, 8: 309-314 (1990) e na publicação WO 92/09690, e uma baixa densidade de revestimento de antígeno, conforme descrito em Marks et al., Biotechnol., 10: 779-783 (1992).
[318] É possível realizar a seleção entre anticorpos de fago de diferentes afinidades para FGFR3, mesmo com afinidades que diferem levemente. Entretanto, mutação aleatória de um anticorpo selecionado (por exemplo, conforme realizado em algumas das técnicas de maturação por afinidade) é susceptível a gerar diversos mutantes, a maioria se ligando ao antígeno, e poucos com alta afinidade. Com a restrição de FGFR3, os raros fagos com alta afinidade podem estar fora da competição (competed out). Para reter todos os mutantes de alta afinidade, os fagos podem ser incubados com excesso de FGFR3 biotinilado, mas com o FGFR3 biotinilado a uma concentração de molaridade mais baixa que a constante de afinidade molar alvo para FGFR3. Os fagos de ligação de alta afinidade podem então ser capturados por esferas paramagnéticas revestidas com estreptoavidina. Esta “captura de equilíbrio” permitem que os anticorpos sejam selecionados de acordo com suas afinidades de ligação, com uma sensibilidade que permite o isolamento de clones mutantes com uma afinidade tão pequena quanto duas vezes maior a partir de um grande excesso de fagos com afinidade mais baixa. As condições utilizadas na lavagem de fagos ligados a uma fase sólida podem também ser manipuladas para discriminar com base nas cinéticas de dissociação.
[319] Os clones FGFR3 podem ser selecionados pela atividade. Em um exemplo de realização, a presente invenção fornece anticorpos FGFR3 que bloqueiam a ligação entre um receptor FGFR3 e seu ligante (tal como FGF1 e/ou FGF9). Clones Fv que correspondem aos ditos anticorpos FGFR3 podem ser selecionados pelo (1) isolamento de clones FGFR3 a partir de uma biblioteca de fago, conforme descrito acima, e opcionalmente submetido a amplificação da população de clones de fago isolada por meio do crescimento da população em um hospedeiro bacteriano apropriado; (2) seleção do FGFR3 e uma segunda proteína contra as quais são desejadas atividades de bloqueio e não bloqueio, respectivamente; (3) adsorção dos clones de fago anti-FGFR3 para imobilizar FGFR3; (4) uso de um excesso da segunda proteína para eluir quaisquer clones indesejados que reconheçam determinantes de ligação de FGFR3 que sobrepõe ou compartilham determinantes de ligação da segunda proteína; e (5) eluição dos clones que permaneceram adsorvidos após a etapa (4). Opcionalmente, os clones com as propriedades de bloqueio/não bloqueio desejadas podem ser adicionalmente enriquecidos por meio da repetição dos procedimentos de seleção descritos uma ou mais vezes no presente.
[320] O DNA que codifica o anticorpo monoclonal derivado do hibridoma ou os clones Fv exibidos por fago da presente invenção são facilmente isolados e sequenciados pelo uso de procedimentos convencionais (por exemplo, pelo uso de primers de oligonucleotídeos desejados para amplificar especificamente as regiões codificantes da cadeia leve e pesada de interesse a partir de um molde (template) de DNA de hibridoma ou fago). Uma vez isolado, o DNA pode ser colocado em vetores de expressão, que são transfectados em seguida para células hospedeiras, tais como células de E. coli, células COS de símios, células de ovário de hamster chinês (CHO) ou células de mieloma que, de outra forma, não produzem proteína de imunoglobulina para obter a síntese de anticorpos monoclonais nas células hospedeiras recombinantes. Artigos de revisão de expressão recombinante em bactérias de DNA que codifica anticorpo incluem Skerra et al., Curr. Opinion in Immunol., 5: 256 (1993) e Pluckthun, Immunol. Revs, 130: 151 (1992).
[321] O DNA que codifica os clones Fv de acordo com a presente invenção pode ser combinado com sequências de DNA conhecidas que codificam regiões constantes de cadeia leve e/ou pesada (por exemplo, as sequências de DNA apropriadas podem ser obtidas a partir de Kabat et al., supra), para formar clones que codificam cadeias leve e/ou pesada, de forma parcial ou integral. Aprecia-se que regiões constantes de qualquer isótipo possam ser utilizadas para este propósito, incluindo regiões constantes de IgG, IgM, IgA, IgD, e IgE, e que estas regiões constantes possam ser obtidas a partir de qualquer espécie animal ou humana. Um clone Fv derivado do DNA de domínio variável de uma espécie animal (tal como humana) e então fundido ao DNA da região constante de outras espécies animais para formar sequências de codificação para “híbrido”, cadeia leve e/ou cadeia pesada de comprimento total é incluída na definição de anticorpo “quimérico” e “híbrido”, conforme utilizado no presente. Em uma realização preferida, um clone Fv derivado do DNA da região variável humana é fundido ao DNA da região constante humana para formar sequência(s) de codificação para cadeias leve e/ou pesada humana de comprimento total ou parcial.
[322] O DNA que codifica o anticorpo anti-FGFR3 derivado de um hibridoma da presente invenção também pode ser modificado, por exemplo, substituindo a sequência de codificação para o domínio constante de cadeia leve e pesada, por sequências homólogas derivadas de clones hibridomas murinos (por exemplo, como no método de Morrison et al., Proc. Natl. Acad. Sci. USA., 81: 6851-6855 (1984)). O DNA que codifica o hibridoma ou o anticorpo derivado do clone Fv ou fragmentos podem ainda ser modificados por junção covalente à sequência codificadora de imunoglobulina na totalidade ou em parte da sequência de codificação para um polipeptídeo não imunoglobulina. Desta forma, anticorpos "quimérico" ou "híbrido" são preparados tendo a especificidade de ligação do clone Fv ou anticorpo derivado do clone do hibridoma da presente invenção.
[323] A presente invenção engloba fragmentos de anticorpos. Em determinadas circunstâncias, há vantagem de se utilizar fragmentos de anticorpos ao invés de anticorpos completos. O menor o tamanho dos fragmentos permite a depuração rápida, e pode levar a um melhor acesso aos tumores sólidos.
[324] Foram desenvolvidas diversas técnicas para a produção de fragmentos de anticorpos. Tradicionalmente, estes fragmentos foram derivados por meio da digestão proteolítica de anticorpos intactos (vide, por exemplo, Morimoto et al., Journal of Biochemical and Biophysical Methods 24:107-117 (1992); e Brennan et al., Science 229:81 (1985)). Entretanto, esses fragmentos podem ser agora produzidos diretamente por células hospedeiras recombinantes. Os fragmentos Fab, Fv e ScFv podem ser todos expressos e secretados em E. coli e assim permitindo a produção de grandes quantidades destes fragmentos. Os fragmentos de anticorpos podem ser isolados a partir das bibliotecas de fagos de anticorpos discutidas acima. Alternativamente, fragmentos Fab’-SH podem ser recuperados diretamente de E. coli e acoplados quimicamente para formar fragmentos F(ab’)2 (Carter et al., Bio/Technology 10:163-167 (1992)). De acordo com outra abordagem, fragmentos F(ab’)2 podem ser isolados diretamente a partir da cultura de células hospedeiras recombinantes. Os fragmentos Fab e F(ab’)2 com maior meia-vida in vivo compreendendo resíduos de epítopos de ligação de receptores recuperados são descritos na patente US 5.869.046. Outras técnicas para a produção de fragmentos de anticorpos serão evidentes para os técnicos no assunto. Em outras realizações, o anticorpo de escolha é um fragmento Fv de cadeia única (scFv) (vide, por exemplo, a publicação WO 93/16185 e as Patentes US 5.571.894 e US 5.587.458). O Fv e sFv são as únicas espécies com sítios de combinação intactos que são desprovidos de regiões constantes; por isso, elas são adequadas para a ligação não especifica reduzida durante a utilização in vivo. As proteínas de fusão sFv podem ser construídas para gerar a fusão de uma proteína efetora na extremidade amino- ou carbóxi- terminal de um sFv. Vide Antibody Engineering, ed. Borrebaeck, supra. O fragmento de anticorpo pode ser também um “anticorpo linear”, por exemplo, conforme descrito na Patente US 5.641.870. Tais fragmentos de anticorpos lineares podem ser monoespecíficos ou biespecíficos.
[325] A presente invenção abrange anticorpos humanizados. Vários métodos de humanização de anticorpos não-humanos são conhecidos no estado da técnica. Por exemplo, um anticorpo humanizado pode conter um ou mais resíduos de aminoácidos nele introduzidos a partir de uma fonte que não é humana. Esses resíduos de aminoácidos não-humanos são muitas vezes denominados resíduos “importados”, que são tipicamente retirados de um domínio variável “importado”. A humanização pode ser essencialmente realizada seguindo-se o método de Winter e colaboradores (Jones et al., Nature, 321:522525 (1986); Riechmann et al., Nature, 332:323-327 (1988); Verhoeyen et al., Science, 239:1534-1536 (1988)), por meio da substituição de sequências de regiões hipervariáveis pelas sequências correspondentes de um anticorpo humano. Consequentemente, tais anticorpos “humanizados” são anticorpos quiméricos (US 4.816.567), em que substancialmente menos de um domínio variável humano intacto foi substituído pela sequência correspondente de uma espécie não humana. Na prática, os anticorpos humanizados são tipicamente anticorpos humanos, em que alguns resíduos da região hipervariável e, possivelmente, alguns resíduos de FR são substituídos por resíduos de sítios análogos em anticorpos de roedores.
[326] A seleção de domínios variáveis humanos, tanto leves quanto pesados, a serem utilizados na fabricação dos anticorpos humanizados é muito importante para reduzir a antigenicidade. De acordo com o então chamado método de “melhor adequação”, a sequência do domínio variável de um anticorpo de roedor é selecionada em comparação com toda a biblioteca de sequências de domínio variável humano conhecido. A sequência humana que está mais próxima da sequência do roedor é então aceita como a região estrutural humana para o anticorpo humanizado (Sims et al. (1993) J. Immunol., 151:2296; Chothia et al. (1987) J. Mol. Biol., 196:901). Outro método utiliza uma região estrutural específica derivada da sequência de consenso de todos os anticorpos humanos de um subgrupo específico de cadeias leve ou pesada. A mesma região estrutural pode ser utilizada para vários anticorpos humanizados diferentes (Carter et al. (1992) Proc. Natl. Acad. Sci. USA, 89:4285; Presta et al. (1993) J. Immunol., 151:2623).
[327] É adicionalmente importante que os anticorpos sejam humanizados com retenção de alta afinidade para o antígeno e outras propriedades biológicas favoráveis. Para atingir este objetivo, de acordo com um método, os anticorpos humanizados são preparados por meio de um processo de análise das sequências parentais e diversos produtos humanizados conceituais, utilizando modelos tridimensionais das sequências parental e humanizada. Modelos de imunoglobulina tridimensionais estão comumente disponíveis e são familiares para os técnicos do assunto. Programas de computador estão disponíveis e mostram e ilustram a provável estrutura conformacional tridimensional de sequências de imunoglobulina candidatas selecionadas. A inspeção dessas exibições permite a análise da provável função dos resíduos no funcionamento da possível sequência de imunoglobulina, isto é, a análise de resíduos que influenciam a capacidade da imunoglobulina candidata de se ligar ao seu antígeno. Desta forma, os resíduos FR podem ser selecionados e combinados a partir das sequências receptora e importada, de forma que seja atingida a característica de anticorpo desejada, tal como maior afinidade para o(s) antígeno(s) alvo(s). Geralmente, os resíduos da região hipervariável estão diretamente e mais substancialmente envolvidos na influência de ligação aos antígenos.
[328] Anticorpos anti-FGFR3 humanos da presente invenção podem ser construídos pela combinação da(s) sequência(s) do domínio variável do clone Fv selecionado a partir de uma biblioteca de exposição por fagos de origem humana com a(s) sequência(s) do domínio constante conhecida(s), conforme descrito acima. Alternativamente, os anticorpos monoclonais anti- FGFR3 humanos da presente invenção podem ser preparados por meio do método de hibridoma. Linhagens celulares de mieloma humano e heteromieloma humano/camundongo para a produção de anticorpos monoclonais humanos foram descritos, por exemplo, por Kozbor, J. Immunol. 133, 3001 (1984), eBrodeur et al., Monoclonal Antibody Production Techniques and Applications, págs. 51-63 (Marcel Dekker, Inc., Nova Iorque, Estados Unidos, 1987) e Boerner et al., J. Immunol., 147: 86 (1991).
[329] É agora possível produzir animais transgênicos (por exemplo, camundongos) que são capazes, mediante imunização, de produzir um repertório completo de anticorpos humanos na ausência de produção de imunoglobulina endógena. Descreveu-se, por exemplo, que a deleção homozigótica do gene da região de ligação de cadeia pesada de anticorpos (JH) em camundongos quiméricos e mutantes de linhagem germinativa resulta na complete inibição da produção de anticorpos endógenos. A transferência do conjunto de genes de imunoglobulina da linhagem germinativa humana em tais linhagens germinativas resultará na produção de anticorpos humanos mediante desafio de antígenos. Vide, por exemplo, Jakobovits et al., Proc. Natl. Acad. Sci. USA, 90:2551 (1993); Jakobovits et al., Nature, 362:255-258 (1993); Bruggermann et al., Year in Immuno., 7:33 (1993).
[330] A transferência de genes pode também ser utilizada para derivar anticorpos a partir de anticorpos não humanos, por exemplo, de roedores, em que o anticorpo humano possui afinidades e especificidades similares ao do anticorpo não humano inicial. De acordo com este método, que também é denominado “impressão de epítopos”, o gene da região variável de anticorpos não humanos tanto de cadeia leve como de cadeia pesada obtidos por meio da técnica de exibição de fagos, conforme descrito acima, é substituído com um repertório de genes de domínio V humanos, criando uma população de scFv ou Fab quiméricos com cadeia não humana/cadeia humana. A seleção com o antígeno resulta no isolamento de um Fab ou scFv quiméricos com cadeia não humana/cadeia humana onde a cadeia humana restaura o sítio de ligação de antígenos destruído, ou seja, o epítopo regula (imprime) a escolha da cadeia humana parceira. Quando o processo for repetido, a fim de substituir a cadeia não humana remanescente, um anticorpo humano é obtido (vide publicação WO 93/06213, publicado em 1 de abril de 1993). Ao contrário da humanização tradicional de anticorpos não humano por enxerto de CDR, esta técnica fornece anticorpos completamente humanos, que não possuem resíduos de CDR ou FR de origem não humana.
[331] Os anticorpos biespecíficos são anticorpos monoclonais, preferencialmente humanos ou humanizados, que possuem especificidades de ligação para pelo menos dois antígenos diferentes. No presente caso, uma das especificidades de ligação é para FGFR3 e a outra é para qualquer outro antígeno. Anticorpos biespecíficos exemplares podem se ligar a dois epítopos diferentes do FGFR3. Os anticorpos biespecíficos podem também ser usados para concentrar agentes citotóxicos nas células que expressam FGFR3. Estes anticorpos possuem um braço de ligação ao FGFR3 e um braço que se liga ao agente citotóxico (por exemplo, saporina, anti-interferon-α, alcalóide de vinca, cadeia A de ricina, metotrexato ou hapteno marcado com isótopo radioativo). Os anticorpos biespecíficos podem ser preparados como anticorpos de comprimento total ou fragmentos destes (por exemplo, anticorpos biespecíficos na forma F(ab')2).
[332] Os métodos de fabricação de anticorpos biespecíficos são conhecidos na técnica. Tradicionalmente, a produção recombinante de anticorpos biespecíficos é baseada na co-expressão de dois pares de cadeia leve e cadeia pesada de imunoglobulina, em que as duas cadeias pesadas possuem especificidades diferentes (Milstein e Cuello, Nature, 305:537-539 (1983)). Devido a classificação aleatória de cadeias pesadas e leves de imunoglobulinas, ditos hibridomas (quadromas) produzem uma mistura potencial de 10 moléculas de anticorpo diferentes, das quais apenas uma possui a estrutura biespecífica correta. A purificação da molécula correta, que normalmente é realizada através de etapas de cromatografia de afinidade, é um tanto problemática e os rendimentos de produto são baixos. Procedimentos similares são descritos na publicação WO 93/08829 publicado em 13 de maio de 1993, e em Traunecker et al., EMBO J., 10:3655 (1991).
[333] De acordo com uma abordagem diferente e, de maior preferência, domínios variáveis de anticorpos com as especificidades de ligação desejadas (sítios de combinação de antígeno e anticorpo) são fundidos à sequências de domínios constantes de imunoglobulina. A fusão está preferivelmente com um domínio constante de cadeia pesada de imunoglobulina, compreendendo pelo menos parte das regiões de dobradiça, CH2 e CH3. Prefere-se ter a primeira região constante de cadeia pesada (CH1) contendo o sítio necessário para ligação de cadeia leve presente em pelo menos uma das fusões. Os DNAs que codificam as fusões de cadeia pesada de imunoglobulina e, se desejado, a cadeia leve de imunoglobulina, são inseridos em vetores de expressão separados e são co-transfectados em um organismo hospedeiro apropriado. Isso proporciona grande flexibilidade no ajuste das proporções mútuas dos 3 fragmentos de polipeptídeos em realizações quando razões desiguais das três cadeias de polipeptídeos utilizadas na construção fornecem os rendimentos ideais. É possível, entretanto, inserir as sequências de codificação para 2 ou todas as 3 cadeias de polipeptídeos em um vetor de expressão quando a expressão de pelo menos duas cadeias de polipeptídeos em razões iguais resulta em alto rendimento ou quando as razões não foram de nenhuma significância específica.
[334] Em um exemplo de realização da presente abordagem, os anticorpos biespecíficos são compostos de uma cadeia pesada de imunoglobulina híbrida com uma primeira especificidade de ligação em um braço, e um par de cadeias leve e pesada de imunoglobulina híbrida (que fornece uma segunda especificidade de ligação) no outro braço. Descobriu-se que essa estrutura assimétrica facilita a separação do composto biespecífico desejado a partir de combinações de cadeia de imunoglobulina indesejadas, pois a presença de uma cadeia leve de imunoglobulina em apenas metade da molécula biespecífica proporciona uma forma fácil de separação. Esta abordagem é descrita na publicação WO 94/04690. Para mais detalhes de produção de anticorpos biespecíficos vide, por exemplo, Suresh et al., Methods in Enzymology, 121:210 (1986).
[335] De acordo com outra abordagem, a interface entre um par de moléculas de anticorpos pode ser elaborada para maximizar o percentual de heterodímeros que são recuperados da cultura de célula recombinante. A interface preferida compreende pelo menos uma parte do domínio CH3 de um anticorpo de domínio constante. Neste método, uma ou mais das cadeias laterais de aminoácidos pequenas da interface da primeira molécula de anticorpo são substituídas por cadeias laterais maiores (por exemplo, tirosina ou triptofano). “Cavidades” compensatórias de tamanhos similares ou idênticos para cadeia(s) lateral(is) grande(s) são criadas na interface da segunda molécula de anticorpo através da substituição de cadeias laterais de aminoácidos grandes com cadeias menores (tal como alanina ou treonina). Esta realização fornece um mecanismo para o aumento no rendimento do heterodímero sobre outros produtos finais indesejados, tais como homodímeros.
[336] Anticorpos biespecíficos incluem anticorpos“heteroconjugados” ou reticulados. Por exemplo, um dos anticorpos no heteroconjugado pode ser acoplado a avidina, e outro a biotina. Tais anticorpos, por exemplo, foram propostos para dirigir células do sistema imune a células indesejadas (US 4.676.980), e para o tratamento de infecção por HIV (WO 91/00360, WO 92/00373, e EP 03089). Anticorpos heteroconjugados podem ser fabricados através do uso de quaisquer métodos convenientes de reticulação. Agentes de reticulação apropriados são conhecidos no estado da técnica, e são descritos, por exemplo, na patente US 4.676.980, dentre uma série de técnicas de reticulação.
[337] Técnicas para a geração de anticorpos biespecíficos a partir de fragmentos de anticorpos também foram descritas na literatura. Anticorpos biespecíficos podem ser preparados, por exemplo, utilizando ligações químicas. Brennan et al., Science, 229:81 (1985) descrevem um procedimento em que anticorpos intactos são clivados proteoliticamente para gerar fragmentos F(ab’)2. Estes fragmentos são reduzidos na presença do agente complexante de ditiol arsenito de sódio para estabilizar ditióis vicinais e evitar a formação de dissulfeto intermolecular. Os fragmentos Fab’ gerados são, então, convertidos em derivados de tionitrobenzoato (TNB). Um dos derivados de Fab’-TNB é então reconvertido no Fab’-tiol por meio da redução com mercaptoetilamina e é misturado com uma quantidade equimolar do outro derivado de Fab’-TNB para formar o anticorpo biespecífico. Os anticorpos biespecíficos produzidos podem ser utilizados como agentes para a imobilização seletiva de enzimas.
[338] Progressos recentes possibilitaram a recuperação direta dos fragmentos Fab’-SH de E. coli, que podem ser acoplados quimicamente para formar anticorpos biespecíficos. Shalaby et al., J. Exp. Med., 175: 217-225 (1992) descrevem a produção de uma molécula F(ab’)2 de anticorpo biespecífico totalmente humanizado. Cada fragmento Fab’ foi secretado separadamente a partir da E. coli e submetido ao acoplamento químico direcionado in vitro para formar o anticorpo biespecífico. O anticorpo biespecífico assim formado foi capaz de ligar-se a células que superexpressam o receptor HER2 e células T humanas normais, bem como iniciar a atividade lítica de linfócitos citotóxicos humanos contra alvos de tumor de mama humano.
[339] Diversas técnicas de produção e isolamento de fragmentos de anticorpos biespecíficos diretamente a partir de cultura de células recombinante também foram descritas. Anticorpos biespecíficos foram produzidos, por exemplo, utilizando zíperes de leucina. Kostelny et al., J. Immunol., 148(5): 1547-1553 (1992). Os peptídeos de zíper de leucina das proteínas Fos e Jun foram ligados às porções Fab’ de dois anticorpos diferentes por meio de fusão gênica. Os homodímeros de anticorpos foram reduzidos na região de dobradiça para formar monômeros e, em seguida, novamente oxidados para formar os heterodímeros de anticorpos. Este método pode também ser utilizado para a produção de homodímeros de anticorpos. A tecnologia de “diacorpo” descrita por Hollinger et al., Proc. Natl. Acad. Sci. USA, 90: 6444-6448 (1993) forneceu um mecanismo alternativo para a fabricação de fragmentos de anticorpos biespecíficos. Os fragmentos compreendem um domínio variável de cadeia pesada (VH) conectado a um domínio variável de cadeia leve (VL) por um ligante que é curto demais para permitir o emparelhamento entre os dois domínios sobre a mesma cadeia. Consequentemente, os domínios VH e VL de um fragmento são forçados a emparelhar-se com os domínios VL e VH complementares de outro fragmento, de maneira a formar dois sítios de ligação de antígenos. Também foi relatada outra estratégia para a fabricação de fragmentos de anticorpos biespecíficos utilizando dímeros Fv de cadeia simples (sFv). Vide Gruber et al., J. Immunol., 152: 5368 (1994).
[340] Anticorpos com mais de duas valências também são contemplados. Por exemplo, anticorpos triespecíficos podem ser preparados. Tutt et al., J. Immunol. 147: 60 (1991).
[341] Um anticorpo multivalente pode ser internalizado (e/ou catabolizado) mais rápido do que um anticorpo bivalente por uma célula que expressa um antígeno para o qual os anticorpos se ligam. Os anticorpos da presente invenção podem ser anticorpos multivalentes (que são diferentes daqueles da classe IgM), com três ou mais sítios de ligação ao antígeno (por exemplo, anticorpos tetravalentes), que podem ser facilmente produzidos pela expressão recombinante de ácidos nucleicos que codificam as cadeias polipeptídicas do anticorpo. O anticorpo multivalente pode incluir um domínio de dimerização e três ou mais sítios de ligação ao antígeno. O domínio de dimerização preferido compreende (ou consiste em) uma região Fc ou uma região de dobradiça. Neste cenário, o anticorpo compreenderá uma região Fc, e três ou mais sítios amino-terminais de ligação do antígeno a região Fc. O anticorpo multivalente preferido na presente invenção compreende (ou consiste em) três a oito, mas preferencialmente, quatro sítios de ligação antigênicos. O anticorpo multivalente compreende pelo menos uma cadeia polipeptídica (e preferencialmente duas cadeias polipeptídicas), onde a(s) cadeia(s) polipeptídica(s) compreende(m) dois ou mais domínios variáveis. Por exemplo, a(s) cadeia(s) polipeptídica(s) pode(m) conter VD1-(X1)n-VD2-(X2)n-Fc, onde VD1 é um primeiro domínio variável, VD2 é um segundo domínio variável, Fc é uma cadeia polipeptídica de uma região Fc, X1 e X2 representam um aminoácido ou polipeptídeo, e n é 0 ou 1. Por exemplo, a(s) cadeia(s) polipeptídica(s) pode(m) compreender: a cadeia da região VH-CH1-ligante flexível-VH-CH1-Fc, ou cadeia região VH-CH1-VH-CH1-Fc. O anticorpo multivalente na presente invenção, preferencialmente compreende ainda de pelo menos dois (e preferencialmente quatro) polipeptídeos de domínio variável de cadeia leve. O anticorpo multivalente da presente invenção pode, por exemplo, compreender entre cerca de dois a cerca de oito polipeptídeos do domínio variável de cadeia leve.
[342] Os polipeptídeos do domínio variável de cadeia leve contemplados na presente invenção compreendem um domínio variável de cadeia leve e compreendem, opcionalmente, um domínio CL.
[343] Em alguns exemplos, a(s) modificação(ões) na(s) sequência(s) de aminoácidos dos anticorpos descritos na presente invenção é(são) contemplada(s). Por exemplo, pode ser desejável melhorar a afinidade de ligação e/ou outras propriedades biológicas do anticorpo. As sequências de aminoácidos variantes do anticorpo são preparadas pela introdução de alterações nucleotídicas apropriadas no ácido nucleico do anticorpo, ou pela síntese peptídica. Essas modificações incluem, por exemplo, deleções e/ou inserções e/ou substituições de resíduos dentro das sequências de aminoácidos do anticorpo. Qualquer combinação de exclusão, inserção e substituição é feita para chegar à construção final, desde que a construção final possua as características desejadas. As alterações nos aminoácidos podem ser introduzidas na sequência de aminoácidos do anticorpo em questão no momento em que sequência é feita
[344] Um método útil para a identificação de determinados resíduos ou regiões do anticorpo que são locais preferidos para mutagênese é denominado “mutagênese de varredura de alanina”, conforme descrito por Cunningham e Wells, (1989) Science, 244: 1081-1085. Na presente invenção, um resíduo alvo ou grupo de resíduos alvos é identificado (por exemplo, resíduos carregados, tais como arg, asp, his, lis e glu) e substituído por um aminoácido neutro ou negativamente carregado (preferivelmente alanina ou polialanina) para afetar a interação dos aminoácidos com antígeno. Esses locais de aminoácidos que demonstram sensibilidade funcional às substituições são então refinados pela introdução de outras variantes ou variantes adicionais nos sítios, ou para os sítios, de substituição. Desta forma, enquanto o sítio para introdução de uma variação de sequência de aminoácidos é predeterminado, a natureza intrínseca da mutação não necessita ser predeterminada. Por exemplo, para analisar o desempenho de uma mutação em um dado sítio, a varredura de ala ou a mutagênese aleatória é conduzida no códon ou região alvo e as imunoglobulinas expressas são selecionadas pela atividade desejada.
[345] As inserções de sequências de aminoácidos incluem fusões de carbóxi e/ou amino-terminais que variam de comprimento desde um resíduo, até polipeptídeos que possuem cem ou mais resíduos, bem como inserções intra-sequenciais de resíduos de aminoácidos únicos ou múltiplos. Exemplos de inserções terminais incluem um anticorpo com um resíduo metionil N-terminal ou anticorpo fundido a um polipeptídeo citotóxico. Outras variantes de inserção da molécula de anticorpo incluem a fusão ao N- ou C-terminal do anticorpo à uma enzima (por exemplo, ADEPT) ou um polipeptídeo que aumente a meia vida do anticorpo no soro.
[346] A glicosilação de polipeptídeos é tipicamente tanto N-ligada quanto O-ligada. N-ligada refere-se à ligação da unidade de carboidrato à cadeia lateral de um resíduo de asparagina. As sequências de tripeptídeos asparagina- X-serina e asparagina-X-treonina, em que X é qualquer aminoácido exceto prolina, são as sequencias de reconhecimento para a ligação enzimática da unidade de carboidrato à cadeia lateral de asparagina. Desta forma, a presença de qualquer destas sequências de tripeptídeos no polipeptídeo cria um sítio de glicosilação em potencial. A glicosilação O-ligada refere-se à ligação de um dos açúcares N-acetilgalactosamina, galactose, ou xilose a um hidroxiaminoácido, mais comumente serina ou treonina, embora 5-hidroxiprolina ou 5-hidroxilisina também possam ser utilizados.
[347] A adição de sítios de glicosilação ao anticorpo é acompanhada, de forma conveniente, pela alteração da sequência de aminoácido de modo que esta sequência contenha uma ou mais das sequências de tripeptídeos descritas acima (para sítios de glicosilação N-ligados). A alteração pode também ser feita através da adição de, ou substituição por, um ou mais resíduos de serina ou treonina à sequência do anticorpo ou polipeptídeo original (para sítios de glicosilação O-ligados).
[348] Quando o anticorpo compreende uma região Fc, os carboidratos ligados a estes podem ser alterados. Por exemplo, anticorpos com uma estrutura de carboidrato madura que carece de fucose anexada a uma região Fc do anticorpo são descritos no Pedido de Patente US 2003/0157108 (Presta, L.). Veja também a US 2004/0093621 (Kyowa Hakko Kogyo Co., Ltd). Anticorpos com uma bisecção por N-acetilglicosamina (GlcNAc) nos carboidratos ligados a uma região Fc do anticorpo são referidos na publicação WO 2003/011878, Jean-Mairet et al. e na Patente US 6.602.684, Umana et al. Anticorpos com pelo menos um resíduo de galactose no oligossacarídeo ligado a uma região Fc do anticorpo são descritos na publicação WO 1997/30087, Patel et al. Veja, também, a publicação WO 1998/58964 (Raju, S.) e WO 1999/22764 (Raju, S.) sobre anticorpos com carboidratos alterados anexados à região Fc deste. Vide também US 2005/0123546 (Umana et al.) sobre moléculas de ligação a antígenos com glicosilação modificada.
[349] A variante de glicosilação preferida na presente invenção compreende uma região Fc, em que uma estrutura de carboidrato ligada à região Fc carece de fucose. Essas variantes podem ter a função ADCC melhorada. Opcionalmente, a região Fc compreende uma ou mais substituições de aminoácidos que melhoram ainda mais a função ADCC, por exemplo, substituições nas posições 298, 333, e/ou 334 da região Fc (numeração de resíduos Eu). Exemplos de publicações relacionadas com variantes deanticorpos "defucosilados" ou "com deficiência de fucose" incluem: US 2003/0157108; WO 2000/61739; WO 2001/29246; US 2003/0115614; US2002/0164328; US 2004/0093621; US 2004/0132140; US 2004/0110704; US2004/0110282; US 2004/0109865; WO 2003/085119; WO 2003/084570; WO2005/035586; WO 2005/035778; WO2005/053742; Okazaki et al. J. Mol. Biol. 336:1239-1249 (2004); Yamane-Ohnuki et al. Biotech. Bioeng. 87: 614 (2004). Exemplos de linhagens de células produtoras de anticorpos defucosilados incluem células Lec13 CHO deficiente na proteína de fucosilação (Ripka et al., Arch. Biochem. Biophys., 249:533-545 (1986); pedido de Patente US 2003/0157108 A1, Presta, L. e WO 2004/056312 A1 Adams et al., especialmente no Exemplo 11), e linhagens de células nocautes, tal como as células CHO nocautes para o gene alfa-1,6-fucosiltransferase, FUT8, (Yamane-Ohnuki et al., Biotech. Bioeng., 87:614 (2004)).
[350] Outro tipo de variante é uma variante de substituição de aminoácidos. Essas variantes têm pelo menos um resíduo aminoácido (pelo menos dois, pelo menos três, pelo menos 4 ou mais) na molécula de anticorpo substituído por um resíduo diferente. Os sítios de maior interesse para mutagênese de substituição incluem as regiões hipervariáveis, mas também são contempladas alterações nas FRs. Substituições conservadoras são exibidas na Tabela 1 sob o título de “substituições preferidas”. Se tais substituições resultarem em uma mudança na atividade biológica, neste caso mais mudanças substanciais, denominadas “substituições exemplares” na Tabela 1, ou conforme descrito mais abaixo em referência às classes de aminoácidos, podem ser introduzidas e os produtos selecionados.
[351] Modificações substanciais nas propriedades biológicas doanticorpo são obtidas por meio da seleção de substituições que diferem significativamente em seus efeitos sobre a manutenção (a) da estrutura da cadeia principal de polipeptídeo na área da substituição, por exemplo, na forma de folha ou em conformação helicoidal; (b) da carga ou hidrofobicidade da molécula no sítio desejado; ou (c) do volume da cadeia lateral. Os resíduos de ocorrência natural podem ser divididos em grupos com base nas propriedades comuns da cadeia lateral.Hidrofóbico: norleucina, met, ala, val, leu, ile;hidrofílico neutro: Cis, Ser, Tre, Asn, Gln;ácido: asp, glu;básico: his, lis, arg;resíduos que influenciam na orientação das cadeias: gli, pro; e aromático: trp, tir, fen.
[352] Substituições não conservadoras causarão a substituição de um membro de uma dessas classes por outra classe.
[353] Um tipo particularmente preferido de variante de substituição envolve a substituição de um ou mais resíduos da região hipervariável de um anticorpo parental (por exemplo, anticorpo humano e humanizado). Geralmente, a(s) variante(s) resultante(s) selecionada(s) para o desenvolvimento adicional terá(ão) propriedades biológicas melhoradas com relação ao anticorpo parental do qual eles foram gerados. Uma maneira conveniente para gerar tais variantes de substituição envolve a maturação de afinidade utilizando a exibição de fago. Resumidamente, diversos sítios de regiões hipervariáveis (por exemplo, 6 a 7 sítios) sofrem mutações para gerar todas as substituições amino possíveis em cada sítio. Os anticorpos gerados dessa forma são exibidos a partir de partículas de fago filamentoso como fusões no produto do gene III do M13 embalado dentro de cada partícula. As variantes exibidas por fago são, então, triadas de acordo com a sua atividade biológica (por exemplo, afinidade de ligação) conforme divulgado na presente invenção. A fim de identificar sítios de regiões hipervariáveis candidatos para a modificação, a mutagênese por varredura de alanina pode ser realizada para identificar resíduos de regiões hipervariáveis que contribuem significativamente para a ligação ao antígeno. Alternativamente ou adicionalmente, pode ser benéfico analisar a estrutura cristalina do complexo antígeno-anticorpo para identificar pontos de contato entre o anticorpo e o antígeno. Tais resíduos de contato e resíduos vizinhos são candidatos para a substituição de acordo com as técnicas elaboradas na presente invenção. Uma vez que tais variantes são produzidas, o painel de variantes é submetido à seleção conforme descrito na presente invenção e os anticorpos com propriedades superiores em um ou mais ensaios relevantes podem ser selecionados para desenvolvimento adicional.
[354] Moléculas de ácidos nucleicos que codificam variantes da sequência de aminoácidos do anticorpo são preparadas por meio de uma série de métodos conhecidos no estado da técnica. Estes métodos incluem, mas não se limitam ao isolamento a partir de uma fonte natural (no caso de variantes de sequências de aminoácidos de ocorrência natural) ou preparação por meio de mutagênese mediada por oligonucleotídeo (ou sítio-dirigida), mutagênese por PCR e cassete de mutagênese de uma variante preparada anteriormente ou uma versão não-variante do anticorpo.
[355] Pode ser desejável introduzir uma ou mais modificações de aminoácidos em uma região Fc dos polipeptídeos de imunoglobulina da presente invenção, gerando assim uma região Fc variante. A região Fc variante humana pode compreender uma sequência da região Fc (por exemplo, sequência da região Fc da IgG1, IgG2, IgG3 ou IgG4 humana) compreendendo uma modificação de aminoácido (por exemplo, uma substituição) em um ou mais posições de aminoácidos incluindo o de uma dobradiça de cisteína.
[356] De acordo com esta descrição e os ensinamentos do estado da técnica, contempla-se que em alguns exemplos de realização um anticorpo usado nos métodos da presente invenção pode compreender uma ou mais alterações em comparação com a contraparte homóloga do tipo selvagem, por exemplo, na região Fc. Estes anticorpos, porém, mantém sensivelmente as mesmas características exigidas para a utilidade terapêutica, comparativamente ao seu homólogo do tipo selvagem. Por exemplo, podem ser feitas algumas alterações na região Fc, que alteraram (isto é, ou melhoram ou diminuem) a ligação ao C1q e/ou a citotoxicidade dependente do complemento (CDC), por exemplo, como descrito na publicação WO99/51642. Vide também Duncan e Winter, Nature 322:738-40 (1988); Patente US 5.648.260; Patente US 5.624.821, e publicação WO94/29351 relativas a outros exemplos de região Fc variantes. A publicação WO 00/42072 (Presta) e WO 2004/056312 (Lowman) descrevem variantes de anticorpos com ligação ao FcRs melhorada ou diminuída. O conteúdo dessas publicações de patentes é especificamente incorporado ao presente pela referência. Vide também, por exemplo, Shields et al. J. Biol. Chem. 9(2):6591-6604 (2001). Anticorpos com a meia vida aumentada e ligação ao receptor Fc neonatal (FcRn) melhorada, que são responsáveis pela transferência de IgGs maternos para o feto (Guyer et al., J. Immunol. 117:587 (1976) e Kim et al., J. Immunol. 24:249 (1994)), são descritos na US 2005/0014934A1 (Hinton et al.). Estes anticorpos compreendem uma região Fc, com uma ou mais substituições neste local que melhoram a ligação da região Fc ao FcRn. As variantes de polipeptídeos com sequências de aminoácidos da região Fc alteradas e capacidade de ligação ao C1q aumentada ou diminuída são descritos na Patente US 6.194.551 B1, publicação WO99/51642. O conteúdo dessaspublicações de patentes é especificamente incorporado ao presente pela referência. Vide, também, Idusogie et al. J. Immunol. 164: 4178-4184 (2000).
[357] Os anticorpos da presente invenção podem ainda ser modificados para conter moléculas adicionais não proteináceas que são conhecidas na técnica e prontamente disponíveis. Preferivelmente, as moléculas adequadas para derivatização do anticorpo são polímeros hidrossolúveis. Exemplos não limitantes de polímeros hidrossolúveis incluem, mas não se limitando a, polietilenoglicol (PEG), copolímeros de etileno-glicol/propilenoglicol, carboximetilcelulose, dextrano, álcool polivinílico, policloreto pirrolidona, poli-1, 3-dioxolano, poli-1,3,6-trioxano, copolímero etileno/maleica anidrido, poliaminoácidos (homopolímeros ou copolímeros aleatórios), e dextrano ou poli (n-vinil pirrolidona) polietileno-glicol, homopolímeros de propropileno glicol, co- polímeros de óxido de prolipropileno óxido de etileno, poliálcool polióis (por exemplo, glicerol), álcool polivinílico, e suas misturas. O polietilenoglicol propionaldeído pode ter vantagens no processo de fabricação devido à sua estabilidade na água. O polímero pode ser de qualquer peso molecular, e pode ser ramificada ou sem ramificação. O número de polímeros acoplados por anticorpo pode variar, e se mais de um polímero encontra-se anexo, os polímeros podem ser a mesma molécula ou moléculas diferentes. Em geral, o número e/ou tipo de polímeros utilizado para derivatização pode ser determinado com base em considerações, incluindo, mas não se limitado a, propriedades específicas ou funções do anticorpo de ser melhorado, se os anticorpos derivados serão utilizados no âmbito de uma terapia, etc.TRIAGEM DE ANTICORPOS COM AS PROPRIEDADES DESEJADAS
[358] Os anticorpos da presente invenção podem ser caracterizados por suas propriedades físicas/químicas e funções biológicas por diversos ensaios conhecidos no estado da técnica (alguns dos quais estão divulgados na presente invenção). Em alguns exemplos de realização, os anticorpos são caracterizados por uma ou mais redução ou bloqueio da ligação do FGF (tal como FGF1 e/ou FGF9), redução ou bloqueio da ativação do FGFR3, redução ou bloqueio da sinalização molecular a jusante de FGFR3, interrupção ou bloqueio da ligação do FGFR3 a um ligante (por exemplo, FGF1, FGF9), redução ou bloqueio da dimerização de FGFR3, promoção da formação do FGFR3 monomérico, ligação ao FGFR3 monomérico, e/ou tratamento e/ou prevenção de um tumor, distúrbio da proliferação celular ou de um câncer; e/ou tratamento ou prevenção de uma doença associada com a expressão e/ou atividade de FGFR3 (tal como a expressão e/ou atividade de FGFR3 aumentada). Em alguns exemplos de realização, os anticorpos são selecionados por aumentarem a ativação de FGFR3, aumentarem a sinalização molecular a jusante do FGFR3, por apresentarem atividade apoptótica, regulação negativa de FGFR3 e função efetora (por exemplo, atividade ADCC).
[359] Os anticorpos purificados podem ser ainda caracterizados por uma série de ensaios, incluindo, mas não se limitando ao, sequenciamento N-terminal, análise de aminoácidos, cromatografia líquida de alta pressão por exclusão de tamanho (HPLC) não desnaturante, espectrometria de massa, cromatografia de troca iônica e digestão pela papaína.
[360] Em alguns exemplos de realização da presente invenção, os anticorpos produzidos são analisados quanto a suas atividades biológicas. Em alguns exemplos de realização, os anticorpos da presente invenção são testados quanto a suas atividades de ligação ao antígeno. Os ensaios de ligação ao antígeno que são conhecidos no estado da técnica e podem ser usados no presente, incluem, mas não estão limitados a, qualquer ensaio de ligação competitivo ou direto usando técnicas como Western blots, radioimunoensaio, ELISA (ensaio imunoadsorvente enzima-associado), imunoensaios do tipo “sanduíche”, ensaios de imunoprecipitação, imunoensaios fluorescentes, e imunoensaios com proteína A. Ensaios de ligação ao antígeno e outros ensaios ilustrativos são apresentados a seguir na seção Exemplos.
[361] Se um anticorpo anti-FGFR3 que inibe o crescimento celular é desejado, o anticorpo candidato pode ser testado utilizando ensaios in vitro e/ou in vivo que medem a inibição do crescimento celular. Se um anticorpo anti- FGFR3 que promove ou não a apoptose é desejado, o anticorpo candidato pode ser avaliado em ensaios que mensuram a apoptose. Métodos para a análise do crescimento e/ou proliferação de uma célula cancerosa, ou métodos para determinar a apoptose de células de câncer são bem conhecidos no estado da técnica e alguns estão descritos e exemplificados na presente invenção. Métodos exemplares para a determinação do crescimento celular e/ou proliferação celular e/ou apoptose incluem, por exemplo, ensaio de incorporação de BrdU, MTT, incorporação de [H3]-timidina (por exemplo, ensaio TopCount (PerkinElmer)), ensaios de viabilidade celular (por exemplo, CellTiter-Glo (Promega)), ensaios de fragmentação do DNA, ensaios de ativação da caspase, exclusão de azul de tripan, ensaios que avaliam a morfologia da cromatina e similares.
[362] Em um exemplo de realização, a presente invenção contempla um anticorpo que possui funções efetoras. Em alguns exemplos de realização, são mensuradas as atividades Fc do anticorpo. Um ensaio de citotoxicidade in vitro ou in vivo pode ser conduzido para confirmar a redução / depleção das atividades CDC e/ou ADCC. Por exemplo, ensaios de ligação com o receptor Fc (FcR) podem ser realizados a fim de garantir que o anticorpo não tenha ligação ao FcyR (e, portanto, carece da atividade ADCC), mas mantém a capacidade de ligação ao FcRn. As células primárias para a mediação de ADCC, as células NK, expressam unicamente Fc(RIII, enquanto monócitos expressam Fc(RI, Fc(RII e Fc(RIII. A expressão de FcR em células hematopoiéticas é resumida na tabela 3, página 464 de Ravetch et al., Annu. Rev. Immunol. 9:45792 (1991). Um exemplo de um ensaio in vitro para avaliar a atividade ADCC de uma molécula de interesse é descrito na Patente US 5.500.362 ou US 5.821.337. Um ensaio para detectar a atividade ADCC também é exemplificado no presente. As células efetoras úteis para tais testes incluem células mononucleares de sangue periférico (PBMC) e células Matadoras Naturais (natural killer - NK). Alternativamente ou adicionalmente, a atividade ADCC da molécula de interesse pode ser avaliada in vivo, por exemplo, em um modelo animal tal como descrito em Clynes et al., PNAS (USA) 95:652-656 (1998). Ensaios de ligação de C1q também podem ser realizados para confirmar que o anticorpo é incapaz de se ligar a C1q e, portanto, carece de atividade CDC. Para avaliar a ativação do complemento, pode ser realizado um ensaio de CDC, conforme descrito, por exemplo, em Gazzano-Santoro et al., J. Immunol. Methods 202:163 (1996). As determinações de ligação ao FcRn e depuração (clearence)/meia-vida in vivo também podem ser realizadas utilizando métodos conhecidos no estado da técnica, por exemplo, aqueles descritos na seção Exemplos.
[363] Se for desejado um anticorpo anti-FGFR3 que se liga ao FGFR3 monomérico, o anticorpo candidato pode ser testado em ensaios (por exemplo, ensaios in vitro) que mensuram a ligação ao FGFR3 monomérico e promoção da formação do FGFR3 monomérico. Tais ensaios são conhecidos no estado da técnica e alguns ensaios estão descritos e exemplificados na presente invenção.
[364] Se for desejado um anticorpo anti-FGFR3 que inibe a dimerização de FGFR3, o anticorpo candidato pode ser avaliado por ensaios de dimerização, por exemplo, conforme descrito e exemplificado na presente invenção.
[365] Em alguns exemplos de realização, a função agonista de FGFR3 do anticorpo candidato é determinada. Métodos para avaliação da função ou atividade agonista dos anticorpos para FGFR3 são conhecidos no estado da técnica e alguns também estão descritos e exemplificados na presente invenção.
[366] Em alguns exemplos de realização, a capacidade de um anticorpo FGFR3 para promover a regulação negativa (down-regulation) do receptor FGFR3 é determinada, por exemplo, usando métodos descritos e exemplificados no presente. Em um exemplo de realização, o anticorpo FGFR3 é incubado com células de teste adequadas, por exemplo, linhagem de células de câncer de bexiga (por exemplo, RT112), e após um período adequado de tempo, os lisados celulares são coletados e analisados quanto aos níveis totais de FGFR3. A análise por FACS também pode ser usada para examinar os níveis de receptores FGFR3 na superfície após a incubação com os anticorpos FGFR3 candidatos.VETORES, CÉLULAS HOSPEDEIRAS E MÉTODOS RECOMBINANTES
[367] Para a produção recombinante de um anticorpo da presente invenção, os ácidos nucleicos que codificam este são isolados e inseridos em um vetor de clonagem replicável para clonagem adicional (amplificação do DNA), ou para expressão. O DNA codificador do anticorpo é facilmente isolado e sequenciado utilizando procedimentos convencionais (utilizando, por exemplo, sondas de oligonucleotídeos que são capazes de se ligar especificamente a genes que codificam as cadeias pesada e leve do anticorpo). Muitos vetores estão disponíveis. A escolha do vetor depende em parte da célula hospedeira a ser utilizada. Em geral, as células hospedeiras preferidas são tanto de origem procariótica quanto eucariótica (geralmente de mamíferos). Aprecia-se que regiões constantes de qualquer isótipo possam ser utilizadas para este propósito, incluindo regiões constantes de IgG, IgM, IgA, IgD, e IgE, e que estas regiões constantes possam ser obtidas a partir de qualquer espécie animal ou humana.
[368] As sequências de polinucleotídeos que codificam polipeptídeos componentes do anticorpo da presente invenção podem ser obtidas por meio de técnicas recombinantes. As sequências de polinucleotídeos desejadas podem ser isoladas e sequenciadas a partir de células produtoras de anticorpos, tais como as células de hibridoma. Alternativamente, os polinucleotídeos podem ser sintetizados usando um sintetizador de nucleotídeo ou por técnicas de PCR. Uma vez obtidos, as sequências de codificação dos polipeptídeos são inseridas em um vetor recombinante capaz de replicar e expressar os polinucleotídeos heterólogos nos hospedeiros procarióticos. Muitos vetores que estão disponíveis e são conhecidos no estado da técnica podem ser utilizados para os propósitos da presente invenção. A seleção de um vetor apropriado dependerá principalmente do tamanho dos ácidos nucleicos a serem inseridos no vetor e da célula hospedeira específica que será transformada com o vetor. Cada vetor contém vários componentes, dependendo da sua função (amplificação ou expressão de polinucleotídeo heterólogo, ou ambos) e sua compatibilidade com a célula hospedeira específica em que ele reside. Os componentes do vetor incluem, mas não estão limitados a: uma origem de replicação, um gene marcador de seleção, um promotor, um sítio de ligação do ribossomo (RBS), uma sequência de sinal, a inserção de ácido nucleico heterólogo e uma sequência de término de transcrição.
[369] De modo geral, vetores plasmidiais contendo replicon e sequências de controle que são derivadas de espécies compatíveis com a célula hospedeira são utilizados em conexão com esses hospedeiros. Normalmente o vetor carrega um sítio de replicação, bem como sequências marcadoras que são capazes de fornecer a seleção fenotípica nas células transformadas. Por exemplo, a E. coli é normalmente transformada usando o pBR322, um plasmídeo derivado de uma espécie E. coli. O pBR322 contém genes de resistência que codificam a ampicilina (Amp) e tetraciclina (Tet) e, portanto, fornece meios fáceis para a identificação de células transformadas. O pBR322, seus derivados, ou outros plasmídeos microbianos ou bacteriófagos também podem conter, ou serem modificados para deste modo conter, os promotores que podem ser usadas pelo organismo microbiano para a expressão de proteínas endógenas. Exemplos de derivados do pBR322 utilizados para expressão de anticorpos específicos estão descritos detalhadamente em Carter et al., Patente US 5.648.237.
[370] Além disso, os vetores fagos contendo replicon e sequências controle que são compatíveis com o microrganismo hospedeiro podem ser usados como vetores de transformação em conexão com esses hospedeiros. Por exemplo, o bacteriófago como o XGEM.TM.-11 podem ser utilizados na produção de um vetor recombinante que pode ser usado para transformar células hospedeiras suscetíveis, tal como a E. coli LE392.
[371] O vetor de expressão da presente invenção pode compreender dois ou mais pares de cistron promotor, cada um codificando um dos componentes de polipeptídeos. Um promotor é uma sequência não traduzida reguladora localizada a montante (5') de um cistron que modula sua expressão. Os promotores procarióticos geralmente caem em duas classes, induzíveis e constitutivos. O promotor induzível é um promotor que inicia níveis aumentados de transcrição do cistron sob seu controle em resposta a alterações na condição de cultura, por exemplo, a presença ou ausência de um nutriente ou mudança na temperatura.
[372] Um grande número de promotores reconhecidos por uma série de células hospedeiras potenciais é conhecido. O promotor selecionado pode ser operacionalmente ligado ao cistron de DNA que codifica a cadeia leve ou pesada pela remoção do promotor a partir do DNA fonte por meio da digestão enzimática com enzimas de restrição e inserção da sequência promotora isolada no vetor da presente invenção. A sequência nativa do promotor e de muitos promotores heterólogos pode ser usada para direcionar a amplificação e/ou expressão dos genes alvo. Em alguns exemplos de realização, os promotores heterólogos são utilizados, pois eles geralmente permitem uma maior transcrição e maior rendimento do gene alvo expresso em comparação com o promotor do polipeptídeo alvo nativo.
[373] Os promotores apropriados para utilização com hospedeiros procarióticos incluem o promotor phoA, sistemas promotores de lactose e β- lactamase, um sistema promotor de triptofano (trp) e promotores híbridos, tais como o promotor tac ou o trc. No entanto, outros promotores que são funcionais em bactérias (por exemplo, outros promotores bacterianos ou de fago conhecidos) também são adequados. Suas sequências de nucleotídeos foram publicadas, permitindo assim que um técnico hábil no assunto possa liga-los de maneira operável (operacionalmente ligado) aos cístrons que codificam as cadeias pesadas e leves alvos (Siebenlist et al., (1980) Cell 20: 269) usando ligantes ou adaptadores para suprir qualquer sítio de restrição desejado.
[374] Em um aspecto da presente invenção, cada cistron dentro do vetor recombinante compreende um componente sequência sinal de secreção que direciona a translocação dos polipeptídeos expressos através da membrana. Em geral, a sequência sinal pode ser um componente do vetor, ou pode ser uma parte do DNA do polipeptídeo alvo que é inserida no vetor. A sequência sinal selecionada para os propósitos da presente invenção pode ser aquela que é reconhecida e processada (ou seja, clivada por uma peptidase sinal) pela célula hospedeira. Para células hospedeiras procarióticas que não reconhecem e processam as sequências sinais nativas para os polipeptídeos heterólogos, a sequência sinal é substituída por uma sequência sinal procariótica selecionada, por exemplo, a partir do grupo que consiste em; fosfatase alcalina, penicilinase, Ipp, ou líderes enterotoxina estável ao calor II (STII), LamB, PhoE, PelB, OmpA e MBP. Em um exemplo de realização da presente invenção, as sequências sinais usadas em ambos os cístrons do sistema de expressão são as sequências sinais STII ou variantes desta.
[375] Em outro aspecto, a produção das imunoglobulinas de acordo com a presente invenção pode ocorrer no citoplasma da célula hospedeira e, portanto, não exige a presença de sequências sinais de secreção no interior de cada cístron. A este respeito, as cadeias leve e pesada de imunoglobulinas são expressas, enoveladas e montadas para formar imunoglobulinas funcionais dentro do citoplasma. Determinadas cepas de hospedeiros (por exemplo, E. coli cepa trxB) fornecem condições citoplasmáticas que são favoráveis para a formação da ligação de pontes de bissulfeto, permitindo assim o adequado enovelamento e montagem das subunidades da proteína expressa. Proba e Pluckthun Gene, 159:203 (1995).
[376] As células hospedeiras procarióticas adequadas para a expressão de anticorpos da presente invenção incluem Archaebacteria e Eubacteria, tais como organismos gram- negativos ou gram-positivos. Exemplos de bactérias úteis incluem a Escherichia (por exemplo, E. coli), Bacilli (por exemplo, B. subtilis), Enterobactérias, espécies de Pseudomonas (por exemplo, P. aeruginosa), Salmonella typhimurium, Serratia marcescans, Klebsiella, Proteus, Shigella, Rhizobia, Vitreoscilla ou Paracoccus. Em um exemplo de realização, são usadas células gram-negativas. Em um exemplo de realização, as células de E. coli são usadas como hospedeiros para a presente invenção. Exemplos das cepas de E. coli incluem as cepas W3110 (Bachmann, Cellular and Molecular Biology, vol. 2 (Washington, D.C.: American Society for Microbiology, 1987), págs. 1190-1219; Depósito ATCC N°: 27.325) e seus derivados, incluindo a cepa 33D3 possuindo o genótipo W3110 ΔfhuA (ΔtonA) ptr3 lac Iq lacL8 ΔompTΔ(nmpc-fepE) degP41 kanR (Patente US 5.639.635). Outras cepas e derivados destas, tal como a E. coli 294 (ATCC 31.446), E. coli B, E. coliX 1776 (ATCC 31.537) e E. coli RV308 (ATCC 31.608) também são adequadas. Estes exemplos são ilustrativos e não limitantes. Métodos para a construção de derivados de qualquer uma das bactérias mencionadas acima que possuem genótipo definido são conhecidos no estado da técnica, por exemplo, em Bass et al., Proteins, 8:309-314 (1990). Geralmente é necessário selecionar as bactérias adequadas levando em consideração a replicabilidade do replicon nas células de uma bactéria. Por exemplo, as espécies E. coli, Serratia ou Salmonella podem ser utilizadas como hospedeiras de maneira adequada, quando plasmídeos bem conhecidos, tais como os plasmídeos pBR322, pBR325, pACYC177 ou pKN410 são utilizados para a construção do replicon. Normalmente a célula hospedeira deve secretar quantidades mínimas de enzimas proteolíticas, e inibidores da protease adicionais podem ser incorporados na cultura celular.
[377] As células hospedeiras são transformadas com os vetores de expressão descritos acima e cultivadas em meio nutriente convencional modificado de forma a ser apropriado para a indução dos promotores, seleção dos transformantes ou amplificação dos genes que codificam as sequências desejadas.
[378] Transformação significa a introdução de DNA no hospedeiro procariótico de forma que o DNA seja replicável, quer seja como um elemento extracromossômicos ou como um integrante cromossômico. Dependendo da célula hospedeira utilizada, a transformação é feita utilizando técnicas padrões apropriada para tais células. O tratamento com cálcio empregando cloreto de cálcio é geralmente utilizado para células bacterianas que possuem barreiras substanciais da parede celular. Outro método de transformação emprega o polietileno glicol/DMSO. No entanto, outra técnica usada é a eletroporação.
[379] As células procarióticas utilizadas para produzir os polipeptídeos da presente invenção são cultivadas em meios conhecidos no estado da técnica e são adequadas para cultivo das células hospedeiras selecionadas. Exemplos de meios apropriados incluem caldo lúria (LB) mais os suplementos nutrientes necessários. Em alguns exemplos de realização, os meios também contêm um agente de seleção, selecionado com base na construção do vetor de expressão, para permitir o crescimento seletivo de células procarióticas que contenham o vetor de expressão. Por exemplo, a ampicilina é adicionada ao meio para o crescimento de células que expressam o gene de resistência à ampicilina.
[380] Quaisquer suplementos necessários além de fontes de carbono, nitrogênio e fosfato inorgânico podem também ser incluídos em concentrações apropriadas introduzidas isoladamente ou como uma mistura com outro suplemento ou meio tal como uma fonte de nitrogênio complexa. Opcionalmente, o meio de cultivo pode conter um ou mais agentes redutores selecionados a partir do grupo que consiste em glutationa, cisteína, cistamina, tioglicolato, ditioeritritol e ditiotreitol.
[381] As células hospedeiras procarióticas são cultivadas sob temperaturas apropriadas. Para crescimento da E. coli, por exemplo, a temperatura preferida varia entre cerca de 20 °C a 39 °C, mais preferivelmente entre 25 °C a cerca de 37 °C, e ainda mais preferivelmente cerca de 30 °C. O pH do meio pode ser qualquer pH que entre 5 a 9, dependendo principalmente do organismo hospedeiro. Para a E. coli, o pH é preferencialmente de cerca de 6,8 a 7,4, de preferencialmente é de 7,0.
[382] Se um promotor induzível é utilizado no vetor de expressão da presente invenção, a expressão da proteína é induzida em condições adequadas para a ativação do promotor. Em um aspecto da presente invenção, promotores PhoA são usados para controlar a transcrição dos polipeptídeos. Assim, as células hospedeiras modificadas são cultivadas em um meio limitante de fosfato para a indução. Preferencialmente, o meio limitante de fosfato é o meio C.R.A.P. (vide, por exemplo, Simmons et al., J. Immunol. Methods. (2002), 263:133-147). Uma variedade de outros indutores podem ser utilizada, de acordo com a construção do vetor utilizado, como é conhecido no estado da técnica.
[383] Em um exemplo de realização, os polipeptídeos expressos da presente invenção são secretados e recuperados a partir do periplasma das células hospedeiras. A recuperação da proteína envolve tipicamente o rompimento do micro-organismo, geralmente por meios como o choque osmótico, sonicação ou lise. Após o rompimento das células, fragmentos celulares ou células inteiras podem ser removidos por meio da centrifugação ou filtragem. As proteínas podem ser adicionalmente purificadas, por exemplo, por cromatografia de afinidade em resina. Alternativamente, as proteínas podem ser transportadas para o meio de cultivo e isoladas nele. As células podem ser removidas do cultivo e o sobrenadante da cultura filtrado e concentrado para purificação adicional das proteínas produzidas. Os polipeptídeos expressos podem ser adicionalmente isolados e identificados utilizando métodos comumente conhecidos, tais como a eletroforese em gel de poliacrilamida (PAGE) e ensaio de Western blot.
[384] Em um aspecto da presente invenção, a produção do anticorpo é realizada em grande quantidade por meio de processos de fermentação. Vários procedimentos de fermentação descontínua alimentada em larga escala estão disponíveis para a produção de proteínas recombinantes. As fermentações em larga escala possuem capacidade de pelo menos 1000 litros, preferencialmente de 1000 a 100.000 litros de capacidade. Estes fermentadores utilizam pás agitadoras ou outros meios apropriados para distribuir oxigênio e nutrientes, especialmente glicose (uma fonte de carbono/energia preferida). A fermentação em pequena escala refere-se geralmente a fermentação em um fermentador que possui capacidade volumétrica não maior que cerca de 100 litros e pode variar de cerca de um 1 litro a 100 litros.
[385] No processo de fermentação, a indução da expressão de proteína é tipicamente iniciada após o crescimento das células sob condições adequadas até uma densidade desejada, tal como em uma OD550 de cerca de 180 a 220, no qual as células estão iniciando a fase estacionária. Uma série de indutores pode ser utilizada, de acordo com a construção de vetor empregado, conforme é sabido no estado da técnica e descrito anteriormente. As células podem ser cultivadas por períodos mais curtos antes da indução. As células normalmente são induzidas durante cerca de 12 a 50 horas, embora possa ser utilizado um tempo maior ou menor de indução.
[386] Para melhorar adicionalmente o rendimento de produção e a qualidade dos polipeptídeos da presente invenção, várias condições de fermentação podem ser modificadas. Para melhorar a montagem e enovelamento adequado do anticorpo secretado, por exemplo, vetores adicionais que superexpressam proteínas chaperonas, tais como proteínas Dsb (DsbA, DsbB, DsbC, DsbD e/ou DsbG) ou FkpA (peptidilprolil cis, transisomerase com atividade de chaperona) podem ser utilizados para co- transformar as células hospedeiras procarióticas. Demonstrou-se que as proteínas de chaperona facilitam o enovelamento adequado e solubilidade de proteínas heterólogas produzidas em células hospedeiras bacterianas. Chen et al., (1999) J. Biol. Chem. 274:19601-19605; Georgiou et al., Patente US6.083.715; Georgiou et al., Patente US 6.027.888; Bothmann e Pluckthun (2000) J. Biol. Chem. 275:17100-17105; Ramm e Pluckthun, (2000) J. Biol. Chem. 275:17106-17113; Arie et al., (2001) Mol. Microbiol. 39:199-210.
[387] Para minimizar a proteólise de proteínas heterólogas expressas (especialmente as que são proteoliticamente sensíveis) certas linhagens hospedeiras com deficiência de enzimas proteolíticas podem ser utilizadas para a presente invenção.
[388] Por exemplo, linhagens de células hospedeiras procarióticas podem ser modificadas para efetuar mutação(ões) genética(s) nos genes que codificam proteases bacterianas conhecidas, tais como Protease III, OmpT, DegP, Tsp, TonA, PhoA, Protease I, Protease Mi, Protease V, Protease VI e combinações destas. Algumas linhagens de E. coli com deficiência de protease estão disponíveis e descritas, por exemplo, em Joly et al., (1998) supra; Georgiou et al. Patente US 5.264.365, Georgiou et al. Patente US 5.508.192; Hara et al., Microbial Drug Resistance, 2:63-72 (1996).
[389] Em um exemplo de realização, as cepas de E. coli deficientes de enzimas proteolíticas e transformadas com um ou mais plasmídeos que superexpressam proteínas chaperonas são utilizadas como células hospedeiras no sistema de expressão da presente invenção.
[390] Podem ser empregados métodos padrões de purificação de proteína conhecidos no estado da técnica. Os procedimentos a seguir são exemplos de procedimentos de purificação adequados: fracionamento em uma coluna de imunoafinidade ou de troca iônica, precipitação em etanol, HPLC de fase reversa, cromatografia em sílica ou em uma resina de troca catiônica, como DEAE, cromatofocusação, SDS-PAGE, precipitação em sulfato de amônio, filtração em gel usando, por exemplo, Sephadex G -75.
[391] Em um aspecto a proteína A imobilizada em uma fase sólida é utilizada para a purificação por imunoafinidade dos produtos anticorpos de comprimento total da presente invenção. A proteína é A é uma proteína da parede celular de 41kD do Staphylococcus aureus que se liga com alta afinidade à região Fc de anticorpos. Lindmark et al., (1983) J. Immunol. Meth. 62:1-13. A fase sólida para que a proteína A seja imobilizada é de preferência uma coluna que compreende uma superfície de vidro ou de sílica, mais preferencialmente uma coluna de vidro com poros controlados ou uma coluna de ácido silícico. Em algumas aplicações, a coluna foi revestida com um reagente, tais como glicerol, na tentativa de evitar a aderência não específica de contaminantes.
[392] Como a primeira etapa de purificação, a preparação derivada da cultura de células, conforme descrito acima, é aplicada sobre a Proteína A imobilizada em fase sólida para permitir a ligação específica do anticorpo de interesse à Proteína A. A fase sólida é então lavada para remover os contaminantes ligados de maneira não especifica à fase sólida. Finalmente, o anticorpo de interesse é recuperado da fase sólida por eluição.B. PRODUÇÃO DE ANTICORPOS UTILIZANDO CÉLULAS HOSPEDEIRAS EUCARIÓTICAS
[393] Os componentes vetores, geralmente incluem, mas não estão limitados a um ou mais dos seguintes: uma sequência sinal, uma origem de replicação, um ou mais genes marcadores, um elemento amplificador, um promotor e uma sequência de terminação de transcrição.(1) COMPONENTE SEQUÊNCIA DE SINAL
[394] Um vetor para uso em uma célula hospedeira eucariótica também pode conter uma sequência de sinal ou outro polipeptídeo que tenha um sítio de clivagem específica na extremidade N-terminal da proteína madura ou polipeptídeo de interesse. A sequência sinal heteróloga selecionada, é aquela que é reconhecida e processada (ou seja, clivada por uma peptidase sinal) pela célula hospedeira. Na expressão celular de mamíferos, as sequências sinais de mamíferos bem como líderes secretórios virais, por exemplo, o sinal gD da herpes simplex, estão disponíveis.
[395] O DNA para tal região precursora é ligada no quadro de leitura para a codificação do DNA do anticorpo.(II) ORIGEM DE REPLICAÇÃO
[396] De modo geral, o componente ‘origem de replicação’ não é necessário para vetores de expressão em mamíferos. Por exemplo, a origem SV40 pode normalmente ser usado somente porque ele contém o promotor precoce.(III) COMPONENTE GENE DE SELEÇÃO
[397] Os vetores de expressão e clonagem irão conter um gene de seleção, também denominado como marcador selecionável. Genes de seleção típicos codificam proteínas que (a) conferem resistência a antibióticos ou outras toxinas, por exemplo, ampicilina, neomicina, metotrexato ou tetraciclina, (b) complementam as deficiências auxotróficas, se for o caso; ou (c) fornecem de nutrientes essenciais que não estão disponíveis a partir do meio complexo.
[398] Um exemplo de um esquema de seleção utiliza um medicamento para suspender o crescimento de uma célula hospedeira. Essas células que são transformadas com sucesso com um gene heterólogo produzem uma proteína que confere resistência à droga e, assim, sobrevivem ao regime de seleção. Exemplos de tal seleção dominante utilizam drogas como a neomicina, ácido micofenólico e higromicina.
[399] Outros exemplos de marcadores de seleção adequados para células de mamíferos são aqueles que permitem a identificação de células competentes que receberem os ácidos nucleicos do anticorpo, tal como DHFR, timidina quinase, metalotioneína-I e -II, preferencialmente genes metalotioneína de primatas, adenosina deaminase, ornitina descarboxilase, etc.
[400] Por exemplo, células transformadas com o gene de seleção DHFR são primeiramente identificadas pelo cultivo de todos os transformantes em um meio de cultura que contenha metotrexato (Mtx), um antagonista competitivo do DHFR. Uma célula hospedeira que é adequada quando o DHFR do tipo selvagem é empregado é a linhagem de células de ovário de hamster chinês (CHO) deficientes na atividade da enzima DHFR (por exemplo, ATCC CRL-9096).
[401] Alternativamente, células hospedeiras (particularmente, células hospedeiras do tipo selvagem que contêm DHFR endógeno) transformadas ou co-transformadas com sequências de DNA que codificam um anticorpo, a proteína DHFR do tipo selvagem e outro marcador selecionável tal como aminoglicosídeo 3’-fosfotransferase (APH) podem ser selecionadas através de crescimento celular em um meio que contém um agente de seleção para o marcador selecionável tal como antibiótico aminoglicosídico, por exemplo canamicina, neomicina ou G418. Vide a Patente US 4.965.199.
[402] Os vetores de expressão e clonagem normalmente contêm um promotor que é reconhecido pelo organismo hospedeiro e é operacionalmente ligado ao ácido nucleico que codifica o polipeptídeo. São conhecidas sequências promotoras para eucariontes. Virtualmente todos os genes eucarióticos contêm uma região rica em AT localizada a cerca de 25 a 30 bases a montante (upstream) do sítio onde é iniciada a transcrição. Outra sequência localizada a 70 a 80 bases a montante (upstream) do início da transcrição de muitos genes é uma região CNCAAT onde N pode ser qualquer nucleotídeo. Na extremidade 3’ da maior parte dos genes eucarióticos, encontra- se uma sequência AATAAA que pode ser o sinal para adição da cauda poli-A à extremidade 3’ da sequência de codificação. Todas estas sequências são inseridas adequadamente em vetores de expressão eucarióticos.
[403] A transcrição do polipeptídeo do anticorpo a partir de vetores em células hospedeiras de mamíferos é controlada, por exemplo, por promotores obtidos a partir dos genomas de vírus tais como polioma vírus, vírus da catapora, adenovírus (tal como Adenovírus 2), vírus de papiloma bovino, vírus de sarcoma aviário, citomegalovírus, retrovírus, vírus da hepatite B e Vírus Símio 40 (SV40), a partir de promotores de mamíferos heterólogos, tais como o promotor de actina ou um promotor de imunoglobulina, de promotores de choque térmico, desde que esses promotores sejam compatíveis com os sistemas de células hospedeiras.
[404] Os promotores inicial e final do vírus SV40 são obtidos convenientemente na forma de fragmento de restrição SV40 que também contém a origem viral SV40 de replicação. O promotor inicial imediato do citomegalovírus humano é obtido convenientemente na forma de fragmento de restrição HindIII E. Um sistema de expressão de DNA em hospedeiros mamíferos utilizando o vírus de papiloma bovino como vetor é descrito na Patente US 4.119.446. Uma modificação deste sistema é descrita na Patente US 4.601.978. Alternativamente, a repetição terminal longa de vírus do Sarcoma de Rous pode ser utilizada como promotor.
[405] A transcrição de um DNA codificador do polipeptídeo do anticorpo da presente invenção por eucariontes superiores é frequentemente aumentada pela inserção de uma sequência amplificadora no vetor. Muitas sequências amplificadoras são agora conhecidas de genes de mamíferos (globina, elastase, albumina, a-fetoproteína e insulina). Tipicamente, entretanto, pode-se utilizar um amplificador (enhancer) de um vírus de célula eucariótica. Exemplos incluem o amplificador SV40 sobre o lado posterior (late side) da origem de replicação (bp 100-270), o amplificador anterior ao promotor do citomegalovírus, o amplificador do polioma sobre o lado posterior da origem de replicação, e amplificador do adenovírus. Veja também Yaniv, Nature 297: 17-18 (1982) sobre elementos de promoção para a ativação de promotores eucarióticos. O amplificador pode também sofrer splicing no vetor em posição 5’ ou 3’ para a sequência codificadora do polipeptídeo de anticorpo, mas encontra- se localizado, preferencialmente, em um sítio a 5’ do promotor.
[406] Os vetores de expressão utilizados em células hospedeiras eucarióticas irão conter tipicamente sequências necessárias para o término da transcrição e para a estabilização do mRNA. Tais sequências são comumente disponíveis a partir da extremidade 5', ocasionalmente 3', regiões não traduzidas de DNAs ou cDNAs virais ou eucarióticos. Estas regiões contêm segmentos de nucleotídeos transcritos na forma de fragmentos poliadenilados na porção não traduzida do mRNA codificador de um anticorpo. Um componente de término de transcrição útil é a região de poliadenilação do hormônio de crescimento bovino. Veja a publicação WO 94/11026 e o vetor de expressão nele descrito.(VII) SELEÇÃO E TRANSFORMAÇÃO DE CÉLULAS HOSPEDEIRAS[001] Células hospedeiras adequadas para clonagem ou expressão do DNA nos vetores da presente invenção incluem as células eucarióticas superiores descritas no presente, incluindo células hospedeiras de vertebrados. A propagação de células de vertebrados em cultura (cultura de tecidos) tornou-se um procedimento de rotina. Exemplos de linhagens celulares de hospedeiros mamíferos incluem linhagem CV1 de rim de macaco transformada com SV40 (COS-7, ATCC CRL 1651); linhagem de rim embrionário humano (293 ou células 293 subclonadas para crescimento em cultura de suspensão, Graham et al., J. Gen. Virol. 36: 59 (1977)); células de rim de filhote de hamster (BHK, ATCC CCL 10); células de ovário de hamster chinês/-DHFR (CHO, Urlaub et al., Proc. Natl. Acad. Sci. USA 77: 4216 (1980)); células de Sertoli de camundongo (TM4, Mather, Biol. Reprod. 23: 243-251 (1980)); células de rim de macacos (CV1 ATCC CCL 70); células de rim de macaco verde africano (VERO-76, ATCC CRL1587); células de carcinoma cervical humano (HELA, ATCC CCL 2); células de rim canino (MDCK, ATCC CCL 34); células de rim de rato “buffalo rat” (BRL 3A, ATCC CRL 1442); células de pulmão humano (W138, ATCC CCL 75); células de fígado humano (Hep G2, HB 8065); tumor da mama de camundongo (MMT 060562, ATCC CCL51); células TRI (Mather et al., Annals N.Y. Acad. Sci. 383: 44-68 (1982)); células MRC 5; células FS4; e uma linhagem de hepatoma humano (Hep G2).
[407] As células hospedeiras são transformadas com os vetores de expressão ou de clonagem descritos acima para produção de anticorpos e cultivadas em meio nutriente convencional modificado de forma a ser apropriado para a indução dos promotores, seleção de transformantes ou amplificação dos genes que codificam as sequências desejadas.
[408] As células hospedeiras utilizadas para a produção de um anticorpo dessa presente invenção podem ser cultivadas em uma variedade de meios de cultura. Meios de crescimento comercialmente disponíveis, tais como o meio Ham's F10 (Sigma), Meio Essencial Mínimo (MEM, Sigma), RPMI-1640 (Sigma), e Meio de Eagle modificado da Dulbecco (DMEM, Sigma) são adequados para o cultivo de células hospedeiras. Além disso, qualquer um dos meios descritos em Ham et al., Meth. Enzymol. 58: 44 (1979), Barnes et al., Anal. Biochem. 102: 255 (1980), Patentes US 4.767.704, US 4.657.866, US 4.927.762, US 4.560.655, US 5.122.469 documentos WO 90/03430; WO 87/00195; ou o pedido de Patente US 30.985, podem ser utilizados como meios de cultura para as células hospedeiras. Qualquer dos ditos meios pode ser suplementados conforme necessário com hormônios e/ou outros fatores de crescimento (tal como insulina, transferina, ou fator de crescimento epidérmico), sais (tal como cloreto de sódio, cálcio, magnésio e fosfato), tampões (tal como HEPES), nucleotídeos (tais como adenosina e timidina), antibióticos (tal como a droga GENTAMYCIN®), elementos traços (definidos como compostos inorgânicos usualmente presentes em concentrações finais na faixa micromolar), e glicose ou uma fonte de energia equivalente. Quaisquer outros suplementos necessários podem também ser incluídos em concentrações adequadas que seria conhecida pelos técnicos no assunto. As condições de cultura tais como, temperatura, pH, e similares são aquelas anteriormente utilizadas com a célula hospedeira selecionada para expressão, e será evidente para o técnico hábil no assunto.
[409] Quando técnicas recombinantes são utilizadas, os anticorpos podem ser produzidos intracelularmente ou secretados diretamente no meio. Se o anticorpo é produzido de forma intracelular, como uma primeira etapa, os resíduos particulados, tanto de células hospedeiras quanto fragmentos lisados, são removidos, por exemplo, por centrifugação ou ultra-filtração. Quando o anticorpo é secretado no meio, os sobrenadantes destes sistemas de expressão são, em geral, primeiramente concentrados pelo uso de um filtro de concentração proteica comercialmente disponível, por exemplo, uma unidade de utrafiltração Amicon ou Millipore Pellicon. Um inibidor de protease tal como o PMSF pode ser incluído em qualquer uma das etapas acima para inibir a proteólise e antibióticos podem ser incluídos para prevenir o crescimento de contaminantes indesejados.[001] A composição de anticorpo preparada a partir das células pode ser purificada através do uso, por exemplo, de cromatografia de hidroxilapatita, eletroforese em gel, diálise e cromatografia de afinidade, com cromatografia de afinidade sendo a técnica de purificação preferida. A adequação da proteína A como ligante de afinidade depende da espécie e do isótipo de qualquer domínio Fc de imunoglobulina que esteja presente no anticorpo. A proteína A pode ser utilizada para purificar anticorpos que são baseados nas cadeias pesadas humanas y1, Y2 ou Y4 (Lindmark et al., J. Immunol. Meth. 62:1-13 (1983)). A proteína G é recomendada para todos os isotipos de camundongos e para a Y3 humana (Guss et al., EMBO J. 5:15671575 (1986)). A matriz ao qual o ligante de afinidade é acoplado é mais frequentemente, agarose, mas também outras matrizes estão disponíveis. Matrizes mecanicamente estáveis tais como vidro com poros controlados ou poli(estirenodivinil)benzeno permitem velocidade de fluxo mais rápido e tempo de processamento mais curto do que aqueles que podem ser alcançados com a agarose. Quando o anticorpo compreende um domínio CH3, a resina Bakerbond ABX® (J. T. Baker, Phillipsburg, NJ) é útil para purificação. Outras técnicas para a purificação de proteína tais como fracionamento em uma coluna de troca iônica, precipitação em etanol, HPLC de fase reversa, cromatografia em sílica, cromatografia em heparina SEPHAROSE®, cromatografia em uma resina de troca catiônica ou aniônica (tal como uma coluna de ácido poliaspártico), cromatofocusação, SDS-PAGE, e precipitação em sulfato de amônio também estão disponíveis dependendo do anticorpo a ser recuperado.
[410] Após qualquer(quaisquer) etapa(s) de purificação preliminar, a mistura compreendendo o anticorpo de interesse e contaminante pode ser submetida a cromatografia de interação hidrofóbica em pH baixo utilizando um tampão de eluição com um pH de cerca de 2,5 a 4,5, preferencialmente realizada sob baixas concentrações de sal (tal como, cerca de 0 a 0,25 M de sal).
[411] A presente invenção também fornece imunoconjugado (designado de maneira intercambiável como “conjugado droga-anticorpo” ou “ADC”) compreendendo qualquer um dos anticorpos anti-FGFR3 descrito na presente invenção conjugado a um ou mais agentes citotóxicos, como, por exemplo, um agente quimioterápico, um medicamento, um agente inibidor do crescimento, uma toxina (por exemplo, uma toxina enzimaticamente ativa de origem bacteriana, vegetal, fúngica ou animal, ou fragmentos destas), ou um isótopo radioativo (ou seja, um radioconjugado).
[412] O uso de conjugados droga-anticorpo para a entrega local de agentes citotóxicos ou citostáticos, ou seja, medicamentos que matam ou inibem o crescimento de células tumorais no tratamento do câncer (Syrigos e Epenetos (1999) Anticancer Research 19:605-614; Niculescu-Duvaz e Springer (1997) Adv. Drg. Del. Rev. 26:151-172; Patente US 4.975.278) permitem a entrega da fração droga aos tumores, levando ao acúmulo intracelular nestes tumores, ao passo que a administração sistêmica destes fármacos de maneira não conjugada pode resultar em níveis inaceitáveis de toxicidade para as células normais, bem como para as células tumorais que se deseja eliminar (Baldwin et al., (1986) Lancet (15 de Março de 1986):603-05; Thorpe, (1985) “Antibody Carriers Of Cytotoxic Agents in Cancer Therapy: A Review,” Em: Monoclonal Antibodies '84: Biological And Clinical Applications, A. Pinchera et al. (ed.s), págs. 475-506). Busca-se desse modo a máxima eficácia com o mínimo de toxicidade. Tanto anticorpos policlonais como anticorpos monoclonais foram relatados como úteis nessas estratégias (Rowland et al., (1986) Cancer Immunol. Immunother., 21:183-87). Os medicamentos utilizados nestes métodos incluem a daunomicina, doxorrubicina, metotrexato e vindesina (Rowland et al., (1986) supra). As toxinas usadas nos conjugados anticorpo-toxina incluem toxinas bacterianas, como a toxina da difteria, toxinas vegetais, tal como a ricina, toxinas tipo pequenas moléculas como a geldanamicina (Mandler et al (2000) Jour. of the Nat. Cancer Inst. 92(19):1573-1581; Mandler et al., (2000) Bioorganic & Med. Chem. Letters 10:1025-1028; Mandler et al., (2002) Bioconjugate Chem. 13:786-791), maitansinóides (EP 1391213; Liu et al., (1996) Proc. Natl. Acad. Sci. USA 93:8618-8623), e caliqueamicina (Lode et al., (1998) Cancer Res. 58:2928; Hinman et al., (1993) Cancer Res. 53:3336-3342). As toxinas podem empregar seus efeitos citotóxicos e citostáticos por mecanismos que incluem a ligação a tubulina, ligação ao DNA ou inibição da topoisomerase. Alguns medicamentos citotóxicos tendem a ser inativados ou se tornam menos ativos quando conjugados com grandes anticorpos ou proteínas ligantes de receptor.
[413] ZEVALIN (ibritumomab tiuxetan, Biogen/Idec) é um conjugado anticorpo-radioisótopo composto de um anticorpo monoclonal IgG1 kappa murino dirigido contra ao antígeno CD20 encontrado na superfície de linfócitos normais e malignos e ligado ao radioisótopo In111 ou Y90 ligado por um quelante ligante a uma tiourea (Wiseman et al. (2000) Eur. Jour. Nucl. Med. 27(7):766-77; Wiseman et al (2002) Blood 99(12):4336-42; Witzig et al (2002) J. Clin. Oncol. 20(10):2453-63; Witzig et al (2002) J. Clin. Oncol. 20(15):3262-69). Embora ZEVALIN tenha a atividade contra o linfoma de célula B não Hodgkin (NHL), a administração resulta em citopenias graves e prolongadas na a maioria dos pacientes. MYLOTARG™ (gemtuzumab ozogamicina, WyethPharmaceuticals), um anticorpo conjugado a droga composto de um anticorpo hu CD33 ligado a caliceamicina, foi aprovado em 2000 para o tratamento da leucemia mielóide aguda por injeção (Drugs of the Future (2000) 25(7):686; Patente US 4970198; US 5079233; US 5585089; US 5606040; US 5693762; US 5739116; US 5767285; US 5773001). Cantuzumab mertansina (Immunogen, Inc.), um conjugado anticorpo-droga composto pelo anticorpo huC242 ligado através do ligante SPP a fração maitansinóide da droga, DM1 do bissulfeto, está avançando em experimentações de fase II para o tratamento dos cânceres que expressam CanAg como, por exemplo, pancreático, gástrico entre outros. MLN- 2704 (Millennium Pharm., BZL Biologics, Immunogen Inc.), um conjugado anticorpo-droga composto pelo anticorpo monoclonal específico para antígeno de membrana anti-prostático (PSMA) ligado a fração maitansinóide da droga, DM1, está sob o desenvolvimento para o tratamento potencial de tumores de próstata. Os peptídeos da auristatina, auristatina E (AE) e a monometilauristatina (MMAE), análogos sintéticos da dolastatina, conjugado aos anticorpos monoclonais quimérico cBR96 (específico para o carcinoma de Lewis Y) e cAC10 (específico para o CD30 em malignidades hematológicas) (Doronina et al. (2003) Nature Biotechnology 21(7):778-784) e estão sob desenvolvimento terapêutico.[001] Os agentes quimioterápicos úteis na geração de tais imunoconjugados são descritos no presente (acima). Toxinas enzimaticamente ativas e fragmentos destas que podem ser utilizadas incluem a cadeia A da difteria, fragmentos ativos não-ligantes da toxina diftérica, cadeia A da exotoxina (da Pseudomonas aeruginosa), cadeia A de rícina, cadeia A da abrina, cadeia A da modeccina, alfa-sarcina, proteínas da Aleurites fordii, proteínas diantina, proteínas da Phytolaca americana (PAPI, PAPII e PAP-S), inibidor da Momordica charantia, curcina, crotina, inibidor da Sapaonaria officinalis, gelonina, mitogellina, restrictocina, fenomicina, enomicina e os tricotecenos. Vide, por exemplo, a publicação WO 93/21232 publicado em 28 de outubro de 1993. Uma série de isótopos radioativos está disponível para a produção de anticorpos radioconjugados. Exemplos incluem Bi212, I131, In131, Y90 e Re186. Osconjugados do anticorpo e agente citotóxico podem ser fabricados utilizando uma série de agentes acopladores de proteínas bifuncionais, tais como propionato de N-succinimidil-3-(2-piridilditiol) (SPDP), iminotiolano (IT), derivados bifuncionais de imidoésteres (tais como adipimidato de dimetila HCl), ésteres ativos (tais como suberato de di-succinimidila), aldeídos (tais como glutaraldeído), compostos bis-azido (tais como bis-(p-azidobenzoila)- hexanodiamina), derivados bis-diazônio (tais como bis-(p-diazoniobenzoil)- etilenodiamina), diisocianatos (tais como 2,6-diisocianato de tolueno) e compostos fluorados bis-ativo (tais como 1,5-difluoro-2,4-dinitrobenzeno). Pode- se preparar, por exemplo, uma imunotoxina ricina, conforme descrito em Vitetta et al., Science 238:1098 (1987). Ácido 1-isotiocianatobenzil-3-metildietileno triaminopentaacético marcado por carbono 14 (MX-DTPA) é um exemplo de agente quelante para conjugação de radionucleotídeo ao anticorpo. Vide a publicação WO94/11026.
[414] Conjugados de um anticorpo e uma ou mais toxinas de moléculas pequenas, tais como caliqueamicina, maitansinóides, dolastatinas, aurostatinas, tricoteno e CC1065 e os derivados destas toxinas que também apresentam atividades de toxinas, são contemplados na presente invenção.
[415] Em alguns exemplos de realizações, o imunoconjugado compreende o anticorpo (de comprimento total ou fragmentos) da presente invenção conjugado a uma ou mais moléculas maitansinóides.
[416] Maitansinóides são inibidores mitóticos que agem através da inibição da polimerização de tubulina. A maitansina foi isolada pela primeira vez a partir da Maytenus serrata (patente US 3.896.111). Descobriu-se em seguida que determinados micróbios também produzem maitansinóides, tais como maitansinol e ésteres C-3 de maitansinol (patente US 4.151.042). Maitansinol sintético, seus derivados e análogos são descritos, por exemplo, nas patentes US 4.137.230; US 4.248.870; US 4.256.746; US 4.260.608; US 4.265.814; US 4.294.757; US 4.307.016; US 4.308.268; US 4.308.269; US 4.309.428; US4.313.946; US 4.315.929; US 4.317.821; US 4.322.348; US 4.331.598; US4.361.650; US 4.364.866; US 4.424.219; US 4.450.254; US 4.362.663 e US 4.371.533.
[417] Frações da droga dos maitansinóides são frações atrativas da droga em conjugados droga-anticorpo porque são: (i) relativamente acessíveis para serem preparadas por fermentação ou modificação química, derivatização de produtos da fermentação, (ii) por serem receptivos à derivatização com os grupos funcionais apropriados para a conjugação através de ligações não dissulfeto aos anticorpos, (iii) são estáveis no plasma, e (iv) eficazes contra uma variedade de linhagens de células tumorais. [001] Os imunoconjugados contendo maitansinóides e métodos para a produção dos mesmos estão descritos, por exemplo, nas patentes US 5.208.020, US 5.416.064 e patente EP 0425235 B1, cujos conteúdos são expressamente incorporados ao presente pela referência. Liu et al., Proc. Natl. Acad. Sci. USA 93: 8618-8623 (1996) descreveram imunoconjugados que compreendem um maitansinóide denominado DM1 ligado ao anticorpo monoclonal C242 dirigido o câncer coloretal humano. Foi observado que o conjugado é altamente citotóxico com relação a células cancerígenas do cólon cultivadas e exibiu atividade antitumoral em teste de crescimento tumoral in vivo. Chari et al., Cancer Research 52: 127-131 (1992) descrevem imunoconjugados em que um maitansinóide foi conjugado por meio de uma ligação de dissulfeto ao anticorpo A7 murino que se liga a um antígeno sobre linhagens de células cancerígenas de cólon humano ou a outro anticorpo TA-1 monoclonal murino que liga o oncogene HER-2/neu. A citotoxicidade do conjugado TA.1- maitansinóide foi testado in vitro em células da linhagem humana SK-BR-3 de câncer de mama, que expressa 3 x 105 antígenos de superfície HER-2 por célula. O conjugado da droga conseguiu um grau de citotoxicidade similar a droga maitansinóide livre, que poderia ser aumentada pelo aumento no número de moléculas maitansinóide por a molécula de anticorpo. Os conjugados A7- maitansinóide demonstrou baixa citotoxicidade sistêmica nos camundongos.
[418] Os conjugados anticorpo-maitansinóide são preparados quimicamente ligando um anticorpo a uma molécula de maitansinóide sem diminuir significativamente a atividade biológica do anticorpo ou da molécula maitansinóide. Vide, por exemplo, a Patente US 5.208.020 (cuja invenção é expressamente incorporada ao presente pela referência). Uma média de 3 a 4 moléculas maitansinóide conjugadas por molécula de anticorpo mostrou eficácia na amplificação da citotoxicidade em células alvo sem afetar negativamente a função ou a solubilidade do anticorpo, embora seja esperado que uma molécula de anticorpo/toxina amplifique a citotoxicidade em relação ao uso de um anticorpo nu. Maitansinóide são bem conhecidos no estado da técnica e podem ser sintetizados por técnicas conhecidas ou isolados a partir de fontes naturais. Maitansinóide adequados são descritos, por exemplo, na patente US 5.208.020 e em outras patentes e publicações não patentarias referidas abaixo na presente invenção. Os maitansinóides preferidos são maitansinol e análogos do maitansinol modificados no anel aromático ou em outras posições da molécula de maitansinol, tais como vários ésteres do maitansinol.
[419] Existem diversos grupos de ligação conhecidos no estado da técnica para a fabricação de conjugados de anticorpo-maitansinóide, que incluem, por exemplo, os descritos na patente US 5.208.020 ou EP 0.425.235 B1 e Chari et al., Cancer Research 52: 127-131 (1992) e pedido de Patente US 10/960.602, depositado em 8 de outubro de 2004, cuja invenção é expressamente incorporada ao presente pela referência. Os conjugados anticorpo-maitansinóide contendo o componente ligante SMCC podem ser preparados conforme divulgado no pedido de Patente US 10/960602, depositado em 8 de outubro de 2004. Os grupos de ligação incluem grupos dissulfeto, grupos tioéter, grupos de ácidos lábeis, grupos fotolábeis, grupos lábeis de peptidase ou grupos lábeis de esterase, conforme descrito nas patentes identificadas acima, sendo os grupos dissulfeto e tioéter os preferidos. Grupos de ligação adicionais estão descritos e exemplificados no presente.
[420] Os conjugados do anticorpo e maitansinóide podem ser feitos através da utilização de uma série de agentes acopladores proteicos bifuncionais, tais com propionato de N-succinimidil-3-(2-piridilditio) (SPDP), cicloexano-1-carboxilato de succinimidil-4-(N-maleimidometila) (SMCC),iminotiolano (IT), derivados bifuncionais de imidoésteres (tais como adipimidato de dimetila HCl), ésteres ativos (tais como suberato de disuccinimidila), aldeídos (tais como glutaraldeído), compostos bis-azido (tais como bis-(para azidobenzoil)-hexanodiamina), derivados de bis-diazônio (tais como bis-(para- diazoniobenzoil)-etilenodiamina), diisocianatos (tais como 2,6-diisocianato de tolueno) e compostos de flúor bis-ativo (tais como 1,5-difluoro-2,4- dinitrobenzeno). Os agentes acopladores particularmente preferidos incluem propionato de N-succinimidil-3-(2-piridiltio) (SPDP) (Carlsson et al., Biochem. J. 173: 723-737 (1978)) e pentanoato de N-succinimidil-4-(2-piridiltio) (SPP) para proporcionar ligação dissulfeto.
[421] O ligante pode estar unido à molécula de maitansinóide em diversas posições, dependendo do tipo da ligação. Uma ligação éster pode ser formada, por exemplo, por meio da reação com um grupo hidroxila, utilizando-se técnicas convencionais de acoplamento. A reação pode ocorrer na posição C-3 que possui um grupo hidroxila, na posição C-14 modificada com hidroximetila, na posição C-15 modificada com um grupo hidroxila e na posição C-20 que possui um grupo hidroxila. Em uma realização preferida, a ligação é formada na posição C-3 de maitansinol ou um análogo do maitansinol.II. AURISTATINA E DOLOSTATINA[001] Em alguns exemplos de realização, o imunoconjugado compreende um anticorpo da presente invenção conjugado a dolastatinas ou peptídicos análogos da dolastatinas e derivados destes, e auristatinas (Patentes US 5.635.483; 5.780.588). As dolastatinas e auristatinas mostraram interferir com a dinâmica do microtúbulo, hidrólise de GTP e divisão nuclear e celular (WOyke et al (2001) Antimicrob. Agents and Chemother. 45(12):3580-3584) e possuem atividade anti-câncer (Patente US 5.663.149) e atividade antifúngica (Pettit et al (1998) Antimicrob. Agents Chemother. 42:2961-2965). A molécula de droga dolastatina ou auristatina pode ser unida covalentemente a um anticorpo através da extremidade N-terminal (amino) ou do C-terminal (carboxila) da fração peptídica da droga (publicação WO 02/088172).
[422] Realizações exemplares da auristatina incluem a extremidade N-terminal ligada as frações DE e DF da droga monometilauristatina, divulgadas em “Monomethylvaline Compounds Capable of Conjugation to Ligands”. US 10/983,340, depositado em 5 de novembro de 2004, cuja invenção é expressamente incorporada ao presente pela referência em sua totalidade.
[423] Tipicamente, as frações de droga baseadas em peptídeo podem ser preparadas formando uma ligação de peptídeos entre dois ou mais aminoácidos e/ou fragmentos de peptídeos. Tais ligações de peptídeos podem ser preparadas, por exemplo, de acordo com o método de síntese de fase líquida (vide, E. Schroder e K. Lübke, “ The Peptides ”, volume 1, págs 76-136, 1965, Academic Press) que é bem conhecido no campo da química de peptídeos. As moléculas de droga auristatina/dolastatina podem ser preparadas de acordo com os métodos: das Patentes US 5.635.483; US 5.780.588; Pettit et al (1989) J. Am. Chem. Soc. 111:5463-5465; Pettit et al (1998) Anti-Cancer Drug Design 13:243277; Pettit, GR, et al. Syntesis, 1996, 719-725; e Pettit et al (1996) J. Chem. Soc. Perkin Trans. 1 5:859-863. Vide também Doronina (2003) Nat Biotechnol 21 (7):778-784; "Monomethylvaline Compounds Capable of Conjugation to Ligands", Patente US 10/983340, depositada em 5 de Novembro de 2004, incorporado ao presente pela referência na sua totalidade (divulgando, por exemplo, ligantes e métodos de preparação de compostos monometilvalina como MMAE e MMAF conjugado aos ligantes).
[424] Em alguns exemplos de realizações, o imunoconjugado compreende um anticorpo da presente invenção conjugado a uma ou mais moléculas de caliqueamicina. A família caliqueamicina de antibióticos é capaz de produzir quebras de DNA de fita dupla em concentrações sub-picomolares. Para a preparação de conjugados da família de caliqueamicina, vide as Patentes US 5.712.374, US 5.714.586, US 5.739.116, US 5.767.285, US 5.770.701, US 5.770.710, US 5.773.001 e US 5.877.296 (todas da American Cyanamid Company). Análogos estruturais de caliqueamicina que podem ser utilizados incluem, mas sem limitar-se a, Y1 I, α2I, α3I, N-acetil—Yl I, PSAG e θI1 (Hinman et al., Cancer Research 53: 3336-3342 (1993), Lode et al., Cancer Research 58: 2925-2928 (1998) e as patentes US da American Cyanamid citadas acima). Outra droga antitumoral à qual o anticorpo pode ser conjugado é QFA, que é um antifolato. Tanto caliqueamicina quanto a QFA possuem sítios intracelulares de ação e não atravessam facilmente a membrana plasmática. Portanto, a absorção celular desses agentes através de internalização mediada por anticorpos aumenta grandemente seus efeitos citotóxicos.
[425] Outros agentes antitumorais que podem ser conjugados aos anticorpos da presente invenção incluem BCNU, estreptozotocina, vincristina e 5-fluorouracil, a família de agentes conhecidos coletivamente como complexo LL-E33288 descrito nas patentes US 5.053.394 e US 5.770.710, bem como as esperamicinas (Patente US 5.877.296).
[426] Toxinas enzimaticamente ativas e fragmentos destas que podem ser utilizadas incluem a cadeia A da difteria, fragmentos ativos não- ligantes da toxina diftérica, cadeia A da exotoxina (da Pseudomonas aeruginosa), cadeia A de rícina, cadeia A da abrina, cadeia A da modeccina, alpha-sarcina, proteínas da Aleurites fordii, proteínas diantina, proteínas da Phytolaca americana (PAPI, PAPII e PAP-S), inibidor da Momordica charantia, curcina, crotina, inibidor da Sapaonaria officinalis, gelonina, mitogellina, restrictocina, fenomicina, enomicina e os tricotecenos. Vide, por exemplo, a publicação WO 93/21232 publicado em 28 de outubro de 1993.
[427] A presente invenção contempla adicionalmente um imunoconjugado formado entre um anticorpo e um composto com atividade nucleolítica (por exemplo, uma ribonuclease ou uma endonuclease de DNA, tal como deoxirribonuclease; DNase).
[428] Para a destruição seletiva do tumor, o anticorpo pode conter um átomo altamente radioativo. Uma série de isótopos radioativos está disponível para a produção de anticorpos radioconjugados. Exemplos incluem At211, I131, I125, Y90, Re186, Re188, Sm153, Bi212, P32, Pb212 e isótopos radioativos de Lu. Quando o conjugado for utilizado para diagnóstico, ele pode compreender um átomo radioativo para estudos cintigráficos, por exemplo, tc99m ou I123, ou um marcador de spin para formação de imagem através de ressonância magnética nuclear (NMR) (também conhecida como formação de imagem por ressonância magnética, MRI), tal como iodo-123, iodo-131, índio- 111, flúor-19, carbono-13, nitrogênio-15, oxigênio-17, gadolínio, manganês ou ferro.[001] O radiomarcador ou outros marcadores podem ser incorporados ao conjugado de formas conhecidas. O peptídeo pode ser, por exemplo, biossintetizado ou pode ser sintetizado por síntese química de aminoácidos, utilizando precursores de aminoácidos apropriados, envolvendo, por exemplo, flúor-19 no lugar de hidrogênio. Marcares como tc99m, I123, Re186, Re188 e In111 podem ser ligados no peptídeo por um resíduo de cisteína. O ítrio-90 (Y90) pode ser ligado a um resíduo de lisina. O método IODOGEN (Fraker et al. (1978), Biochem. Biophys. Res. Commun. 80: 49-57) pode ser utilizado para a incorporação de iodo-123. A publicação “Monoclonal Antibodies in Immunoscintigraphy” (Chatal, CRC Press, 1989) descreve outros métodos em detalhes.
[429] Os conjugados do anticorpo e agentes citotóxicos podem ser feitos através da utilização de uma série de agentes acopladores proteicos bifuncionais, tais com propionato de N-succinimidil-3-(2-piridilditio) (SPDP), cicloexano-1-carboxilato de succinimidil-4-(N-maleimidometila) (SMCC), iminotiolano (IT), derivados bifuncionais de imidoésteres (tais como adipimidato de dimetila HCl), ésteres ativos (tais como suberato de disuccinimidila), aldeídos (tais como glutaraldeído), compostos bis-azido (tais como bis-(para- azidobenzoil)-hexanodiamina), derivados de bis-diazônio (tais como bis-(para- diazoniobenzoil)-etilenodiamina), diisocianatos (tais como 2,6-diisocianato de tolueno) e compostos de flúor bis-ativo (tais como 1,5-difluoro-2,4- dinitrobenzeno). Pode-se preparar, por exemplo, uma imunotoxina ricina, conforme descrito em Vitetta et al., Science 238:1098 (1987). Ácido 1-isotiocianatobenzil-3-metildietileno triaminopentaacético marcado por carbono 14 (MX-DTPA) é um exemplo de agente quelante para conjugação de radionucleotídeo ao anticorpo. Vide a publicação WO94/11026. O ligante pode ser um “ligante clivável” que facilita a liberação da droga citotóxica na célula. Pode-se utilizar, por exemplo, um ligante ácido lábil, ligante sensível à peptidase, ligante fotolábel, ligante de dimetila ou ligante contendo dissulfeto (Chari et al., Cancer Research 52: 127-131 (1992); patente US 5.208.020).
[430] Os compostos da presente invenção contemplam expressamente, mas não são limitados a, ADC preparado com reagentes reticulantes: BMPS, EMCS, GMBS, HBVS, LC-SMCC, MBS, MPBH, SBAP, SIA, SIAB, SMCC, SMPB, SMPH, sulfo-EMCS, sulfo-GMBS, sulfo-KMUS, sulfo-MBS, sulfo-SIAB, sulfo-SMCC, sulfo-SMPB, e SVSB (succinimidil-(4-vinilsulfona)benzoato) que são comercialmente disponíveis (por exemplo, pela Pierce Biotechnology, Inc., Rockford, IL., EUA). Veja as páginas 467-498 de Applications Handbook and Catalog 2003-2004.
[431] Nos conjugados de anticorpos com drogas (ADC) da presente invenção, um anticorpo (Ab) é conjugado a uma ou mais frações de droga (D), por exemplo, cerca de 1 a 20 moléculas de droga por anticorpo, por meio de um ligante (L). O ADC de Fórmula I pode ser preparado por diversos protocolos, empregando reações, condições e reagentes de química orgânica conhecidos pelos técnicos hábeis no assunto, incluindo: (1) a reação de um grupo nucleofílico de um anticorpo com um reagente ligante bivalente a forma Ab-L, através de uma ligação covalente, seguida pela reação com a fração da droga D; e (2) reação de um grupo nucleofílico de uma fração da droga com um reagente ligante bivalente para formar D-L, através de uma ligação covalente, seguida pela reação com o grupo nucleofílico de um anticorpo. Métodos adicionais para preparação de ADC estão descritos no presente.
[432] O ligante pode ser composto por um ou mais componentes ligante. Exemplos componentes de ligantes incluem 6-maleimidocaproil (“CM”), maleimidopropanoil (“MP”), valina-citrulina (“val-cit”), alanina-fenilalanina (“alafen”), p-aminobenziloxicarbonil (“PAB”), N-Succinimidil 4-(2-piridiltio) pentanoato (“SPP”), N-Succinimidol 4-(N-maleimidometil) ciclohexano-1 carboxilato (“SMCC”), e Succinimidil N-(4-iodo-acetil) aminobenzoato (“SIAB”).Componentes ligantes adicionais são conhecidos no estado da técnica e alguns estão descritos no presente documento. Vide também "Monomethylvaline Compounds Capable of Conjugation to Ligands", US N° 10/983340, depositado em 5 de Novembro de 2004, cujo conteúdo é incorporado ao presente pela referência em sua totalidade.
[433] Em alguns exemplos de realização, o ligante pode compreender resíduos de aminoácidos. Exemplos de componentes ligantes de aminoácidos incluem dipeptídeos, tripeptídeos, tetrapeptídeos ou pentapeptídeos. Dipeptídeos exemplares incluem: valina-citrulina (vc ou val-cit), fenilalanina-alanina (af ou ala-fen). Tripeptídeos exemplares incluem: glicina- valina-citrulina (gli-val-cit) e glicina-glicina-glicina (gli-gli-gli). Resíduos de aminoácidos que compreendem um componente aminoácido ligante incluem aqueles que ocorrem naturalmente, bem como aminoácidos menores e aminoácidos análogos de ocorrência não natural, como a citrulina. Componentes ligantes de aminoácidos podem ser concebidos e otimizados em sua seletividade para a clivagem enzimática por meio de enzimas específicas, por exemplo, uma protease associada ao tumor, catepsinas B, C e D, ou uma protease plasmina.
[434] Grupos nucleofílicos em anticorpos incluem, mas não estão limitados a: (i) grupos amino N-terminal, (ii) grupos amina da cadeia lateral, por exemplo, lisina, (iii) grupos tióis de cadeia lateral, por exemplo, de cisteína, e (iv) grupos açúcares hidroxila ou amino, em que o anticorpo está glicosilado. Grupos amina, tiol e hidroxila são nucleofílicos e são capazes de reagir para formar ligações covalentes com grupos eletrofílicos nas frações ligantes e reagentes ligantes, incluindo: (i) ésteres de ativos, tais como ésteres NHS, ésteres HOBt, haloformatos e ácidos halogenetos, (ii) haletos de alquila e benzila como haloacetamidas, (iii) aldeídos, cetonas, carboxila e grupos maleimida. Certos anticorpos possuem dissulfetos intercadeias redutíveis, ou seja, pontes de cisteína. Os anticorpos podem ser reativos para a conjugação com os reagentes ligantes pelo tratamento com um agente redutor, tal como o DTT (ditiotreitol). Cada ponte de cisteína forma, teoricamente, dois tiois nucleofílicos reativos. Grupos nucleofílicos adicionais podem ser introduzidos em anticorpos por meio da reação das lisinas com 2-iminotiolano (reagente de Traut), resultando na conversão de uma amina em um tiol. Os grupos tióis reativos podem ser introduzidos no anticorpo pela introdução de um, dois, três, quatro ou mais resíduos de cisteína (por exemplo, preparando anticorpos mutantes compreendendo um ou mais resíduos de aminoácido cisteína não nativos).
[435] O conjugado droga-anticorpo da presente invenção pode também ser produzido pela modificação do anticorpo para introduzir porções eletrofílicas, que podem reagir com substituintes nucleofílicos no reagente ligante ou droga. Os açúcares dos anticorpos glicosilados podem ser oxidados, por exemplo, com reagentes periodato oxidantes, formando grupos aldeído ou cetona que podem reagir com o grupo amina do reagente ligante ou molécula de droga. Os grupos de base de Schiff imina resultantes podem formar uma ligação estável, ou podem ser reduzidos, por exemplo, por meio de reagentes borohidreto para formar ligações amina estáveis. Em um exemplo de realização, a reação da porção carboidrato de um anticorpo glicosilado com qualquer galactose oxidase ou meta-periodato de sódio pode resultar em grupos carbonila (aldeído e cetona) na proteína que pode reagir com grupos apropriados na droga (Hermanson, Bioconjugate Techniques). Em outro exemplo de realização, as proteínas contendo resíduos de serina ou treonina N-terminal podem reagir com o meta-periodato de sódio, resultando na produção de um aldeído no lugar do primeiro aminoácido (Geoghegan & Stroh, (1992) Bioconjugate Chem. 3:138146; Patente US 5.362.852). Tal aldeído pode reagir com uma molécula de droga ou ligante nucleofílico.
[436] Da mesma forma, grupos nucleofílicos na molécula de droga incluem, mas não estão limitados a: Grupos amina, tiol, hidroxila, hidrazida, oxima, tiosemicarbazona hidrazina, carboxilato hidrazina, e arilhidrazida capazes de reagir para formar ligações covalentes com grupos eletrofílicos na frações ligante e reagentes ligante, incluindo: (i) ésteres de ativos, tais como ésteres NHS, ésteres HOBt, haloformatos e ácidos halogenetos, (ii) haletos de alquila e benzila como haloacetamidas, (iii) aldeídos, cetonas, carboxila e grupos maleimida.
[437] Alternativamente, pode ser feita uma fusão proteica compreendendo o anticorpo e o agente citotóxico, por exemplo, por técnicas recombinantes ou pela síntese de peptídeos. O comprimento de DNA pode conter regiões respectivas que codificam as duas porções do conjugado adjacentes ou separadas por uma região que codifica um peptídeo ligante que não destrua as propriedades desejadas do conjugado.
[438] Ainda em outra realização, o anticorpo pode ser conjugado a um “receptor” (tal como estreptavidina) para a utilização no pré-direcionamento tumoral, em que o conjugado de anticorpo-receptor é administrado ao paciente, seguido pela remoção do conjugado não ligado da circulação utilizando um agente de limpeza e, em seguida, um “ligante” (por exemplo, a avidina) que está conjugado a um agente citotóxico (por exemplo, um radionucleotídeo) é administrado.MÉTODOS DE USO DOS ANTICORPOS ANTI-FGFR3
[439] A presente invenção apresenta o uso de um anticorpo FGFR3 como parte de um esquema de tratamento específico destinado a proporcionar um efeito benéfico pela atividade deste agente terapêutico. A presente invenção é particularmente útil no tratamento do câncer de vários tipos em várias fases.
[440] O termo câncer abrange um conjunto de desordens proliferativas, incluindo, mas não limitado ao, crescimento pré-canceroso, tumores benignos e tumores malignos. Os tumores benignos permanecem localizados no local de origem e não possuem a capacidade de infiltrar, invadir, ou realizar metástases para locais distantes. Os tumores malignos irão invadir e danificar outros tecidos ao seu redor. Eles também podem obter a capacidade de romper o local de origem e se espalhar para outras partes do corpo (metástase), geralmente através da corrente sanguínea ou através do sistema linfático, onde os nódulos linfáticos (linfonodos) estão localizados. Os tumores primários são classificados pelo tipo de tecido ao qual surgem; tumores metastáticos são classificados pelo tipo de tecido a partir do qual as células cancerosas são derivadas. Ao longo do tempo, as células de um tumor maligno se tornam mais anormais e se parecem menos como as células normais. Esta mudança na aparência das células cancerosas é chamada de grau do tumor e as células cancerosas são descritas como sendo bem diferenciadas (baixo grau), moderadamente diferenciadas, pouco diferenciadas ou indiferenciadas (alto grau). Células bem diferenciadas são de aparência bastante normal e se assemelham com células normais a partir da qual elas se originaram. Células indiferenciadas são células que se tornaram muito anormais de modo que não é possível determinar a origem das células.
[441] Os sistemas de estadiamento do câncer descrevem o quanto o câncer se espalhou anatomicamente e tentam colocar os pacientes com prognóstico e tratamento similar em grupos de mesmo estágio. Vários testes podem ser realizados para ajudar no estadiamento do câncer, incluindo biópsia e exames de imagem, como radiografia de tórax, mamografia, cintilografia óssea, tomografia computadorizada (CT) e ressonância magnética (MRI). Os exames de sangue e uma avaliação clínica também são utilizados para avaliar a saúde geral do paciente e detectar se o câncer se espalhou para determinados órgãos.
[442] Para o estadiamento do câncer, a American Joint Commitee on Cancer primeiramente classifica os cânceres, particularmente os tumores sólidos, em uma categoria de letras utilizando o sistema de classificação TNM. Os cânceres são designados com a letra T (tamanho do tumor), N (nódulos palpáveis), e/ou M (metástases). T1, T2, T3 e T4 descrevem o aumento de tamanho da lesão primária, N0, N1, N2 e N3 indicam progressivamente o avanço no envolvimento de linfonodos, e M0 e M1 refletem a ausência ou presença de metástases à distância.
[443] Em um segundo método de estadiamento, também conhecido como Overall Stage Grouping ou Roman Numeral Staging, os cânceres são divididos em estágios que vão de 0 a IV, incorporando a dimensão das lesões primárias, bem como a presença de disseminação para linfonodos e metástases à distância. Neste sistema, os casos são agrupados em quatro estágios indicados por algarismos romanos de I a IV, ou são classificados como "recorrente". Para alguns tipos de câncer, o estágio 0 é conhecido como "in situ" ou "Tis", tais como carcinoma ductal in situ ou carcinoma lobular in situ para o câncer de mama. Adenomas de alto grau também podem ser classificados como estágio 0. Em geral, os cânceres de fase I são pequenos cânceres localizados que geralmente são curáveis, enquanto cânceres de estágio IV geralmente representam cânceres inoperáveis ou metastáticos. Os estágios II e III são geralmente cânceres localmente avançados e/ou que apresentam o envolvimento de gânglios linfáticos locais. Em geral, os estágios mais elevados indicam uma doença mais extensa, incluindo grande tamanho tumoral e/ou disseminação do câncer para os linfonodos próximos e/ou órgãos adjacentes ao tumor primário. Estes estágios são definidos com precisão, mas a definição é diferente para cada tipo de câncer e é conhecido pelo técnico do assunto.
[444] Muitos registros de câncer, como a vigilância do instituto nacional do câncer (NCI - National Cancer Institute), e o programa de vigilância e epidemiologia (SEER - Surveillance, Epidemiology, and End Results), sumarizam o uso do estadiamento. Este sistema é utilizado para todos os tipos de cânceres. Ela agrupa os casos de câncer em cinco categorias principais:
[445] In situ é um câncer precoce que está presente apenas na camada de células em ele que começou.
[446] Localizado é um câncer que se limita ao órgão em que começou, sem evidência de propagação.
[447] Regional é o câncer que se espalhou além do local original (primário) para os linfonodos ou órgãos e tecidos vizinhos.
[448] Distante é um câncer que se espalhou a partir do local primário para órgãos distantes ou linfonodos distantes.
[449] Desconhecido é usado para descrever os casos para os quais não existe informação suficiente para indicar um estágio.
[450] Além disso, é comum para o câncer retornar meses ou anos após o tumor primário ser removido. O câncer que recorre após todo o tumor visível ser erradicado, é chamado de doença recorrente. A doença que se repete na área do tumor primário é localmente recorrente e doença que se repete como metástase é denominada como uma recorrente distante.
[451] O tumor pode ser um tumor sólido ou um tumor de tecido não-sólido ou mole. Exemplos de tumores de tecidos moles incluem a leucemia (por exemplo, leucemia mieloide crônica, leucemia mieloide aguda, leucemia linfoide aguda em adultos, leucemia mieloide aguda, leucemia linfoblástica aguda de células-B maduras, leucemia linfocítica crônica, leucemia polimfocítica ou leucemia de células pilosas) ou linfoma (por exemplo, linfoma não-Hodgkin, linfoma cutâneo de células T, ou doença de Hodgkin). Um tumor sólido inclui qualquer tipo de câncer em outros tecidos do corpo que não o sangue, medula óssea ou sistema linfático. Os tumores sólidos podem ainda ser divididos entre aqueles de células de origem epitelial e aqueles de células de origem não- epitelial. Exemplos de células epiteliais de tumores sólidos incluem tumores do trato gastrointestinal, cólon, mama, próstata, pulmão, rim, fígado, pâncreas, ovário, cabeça e pescoço, cavidade oral, estômago, duodeno, intestino delgado, intestino grosso, ânus, vesícula biliar, lábio, nasofaringe, pele, útero, órgãos genitais masculinos, órgãos do aparelho urinário, bexiga e pele. Tumores sólidos, de origem não epitelial incluem sarcomas, tumores cerebrais e tumores ósseos. Outros exemplos de tumores estão descritos na seção Definições.
[452] Em alguns exemplos de realização, o paciente é submetido a um teste diagnóstico, por exemplo, antes e/ou durante e/ou após a terapia. Geralmente, se um exame diagnóstico é realizado, a amostra pode ser obtida de um paciente que necessita de terapia. Se o indivíduo tiver câncer, a amostra pode ser uma amostra do tumor, ou outras amostras biológicas, tais como fluidos biológicos, incluindo, sem se limitar a, sangue, urina, saliva, líquido ascítico, ou derivados destes como o soro do sangue e plasma sanguíneo, e similares.
[453] A amostra biológica no presente pode ser uma amostra fixada, por exemplo, fixada em formalina, amostra emblocada em parafina (FFPE), ou amostra congelada.
[454] Vários métodos para determinar a expressão de mRNA ou proteína incluem, mas não estão limitados a, perfis de expressão gênica, reação em cadeia da polimerase (PCR), incluindo a PCR quantitativa em tempo real (qRT-PCR), análise de microarranjos, a análise serial da expressão gênica (SAGE), MassARRAY, analise da expressão gênica por Sequenciamento de Assinaturas. Massivamente Paralelo (MPSS), proteômica, imuno-histoquímica (IHQ), e etc. Preferencialmente o mRNA é quantificado. Tal análise de mRNA é realizada preferencialmente através da técnica de reação em cadeia da polimerase (PCR), ou pela análise de microarranjo. Quando a PCR é empregada, a forma preferida de PCR é a PCR quantitativa em tempo real (qRT- PCR). Em uma realização, a expressão de um ou mais genes acima observados é considerado a expressão positiva estiver na média ou acima, por exemplo, em comparação com outras amostras do mesmo tipo de tumor. O nível de expressão mediana pode ser determinado, essencialmente, de maneira simultânea com a medição da expressão gênica, ou pode ser determinado anteriormente.
[455] As etapas de um protocolo representativo para o perfil da expressão gênica usando tecidos fixados, emblocados em parafina como fonte de RNA, incluindo o isolamento do mRNA, a purificação, a extensão de primers e amplificação são dadas em vários artigos publicados em revistas (por exemplo: Godfrey et al. J. Molec. Diagnostics 2: 84-91 (2000); Specht et al., Am. J. Pathol. 158: 419-29 (2001)). Resumidamente, um processo representativo começa com o corte de cerca de 10 microgramas de espessura de amostras emblocadas em parafina de tecido do tumor. O RNA é então extraído e as proteínas e o DNA são removidos. Após a análise da concentração de RNA, etapas de reparação e/ou amplificação de RNA podem ser incluídas, se necessário, e RNA é transcrito usando promotores específicos de genes seguido por PCR. Finalmente, os dados são analisados para identificar a melhor opção de tratamento disponível para o paciente, com base no padrão característico da expressão gênica identificada na amostra de tumor analisada.
[456] A detecção do gene ou a expressão da proteína pode ser determinada diretamente ou indiretamente.
[457] Pode-se determinar a expressão ou amplificação de FGFR3 no câncer (direta ou indiretamente). Vários ensaios diagnósticos/prognósticos estão disponíveis para esse fim. Em um exemplo de realização, a superexpressão de FGFR3 pode ser analisada por IHQ. Cortes de tecido emblocados em parafina de uma biópsia de tumor podem ser submetidos ao ensaio de IHQ e atribuindo uma intensidade de coloração para a proteínaFGFR3 seguindo os critérios:
[458] Escore 0 nenhuma marcação ou coloração de membrana é observada em menos de 10% das células tumorais.
[459] Escore 1+ a coloração na membranaescassamente/fracamente perceptível é detectada em mais de 10% das células tumorais. As células são coradas em apenas parte de sua membrana.
[460] Escore 2+ a coloração na membrana de fraco a moderado é detectada em mais de 10% das células tumorais.
[461] Escore 3+ a coloração na membrana de moderado a forte é detectada em toda a membrana em mais de 10% das células tumorais.
[462] Em alguns exemplos de realização, os tumores com escores 0 ou 1 para a avaliação da superexpressão de FGFR3 pode ser caracterizada como uma não superexpressão de FGFR3, enquanto tumores com escores 2 ou 3 podem ser caracterizados como uma superexpressão de FGFR3.
[463] Em alguns exemplos de realização, os tumores com superexpressão de FGFR3 podem ser avaliados por um escore de imunohistoquímica correspondente ao número de cópias de moléculas FGFR3 expressas por célula, e podem ter sido determinadas bioquimicamente:0 = 0-90 cópias/célula, 1+ = pelo menos cerca de 100 cópias/célula,2+ = pelo menos cerca de 1000 cópias/célula,3+ = pelo menos cerca de 10.000 cópias/célula,
[464] Alternativamente, ou adicionalmente, um ensaio FISH pode ser realizado em tecidos de tumores fixados em formalina e emblocados em parafina para determinar a presença e/ou extensão (se houver) da amplificação de FGFR3 no tumor.
[465] A ativação de FGFR3 pode ser determinada diretamente (por exemplo, pelo ensaio de fosfo-ELISA, ou outros métodos para detecção de receptor fosforilado) ou indiretamente (por exemplo, pela detecção de componentes ativados a jusante da via de sinalização, detecção de dímeros de receptores (por exemplo, homodímeros, heterodímeros), detecção de perfis de expressão gênica e técnicas similares).
[466] Da mesma forma, a ativação constitutiva de FGFR3 e/ou ativação de FGFR3 independente de ligante ou dependente de ligante, podem ser detectadas diretamente ou indiretamente (por exemplo, pela detecção de mutações no receptor correlacionado com a atividade constitutiva, pela detecção da amplificação do receptor correlacionado com a atividade constitutiva e etc.).
[467] Métodos para detecção de mutações do ácido nucleico são bem conhecidos no estado da técnica. Muitas vezes, embora não necessariamente, o ácido nucleico alvo em uma amostra é amplificado para fornecer a quantidade desejada de material para determinar se uma mutação está presente. As técnicas de amplificação são bem conhecidas no estado da técnica. Por exemplo, o produto amplificado pode ou não incluir todas assequências de ácido nucleico que codificam a proteína de interesse, desde que o produto amplificado compreenda a posição da sequência deaminoácido/ácidos nucleicos específica, onde a mutação é suspeita de estar.
[468] Em um exemplo, a presença de uma mutação pode ser determinada pelo contato com ácido nucleico de uma amostra com uma sonda de ácido nucleico que é capaz de hibridizar especificamente ao ácido nucleico que codifica um ácido nucleico mutante, e então detectando a dita hibridização. Em uma realização, a sonda é marcada para a detecção, por exemplo, com um radioisótopo (H3, P32, P33, etc.), um agente fluorescente (rodamina,fluoresceína e etc.) ou um agente cromogênico. Em algumas realizações, a sonda é um oligômero antisense, por exemplo, PNA, morfolino-fosforamidatos, LNA ou 2'-alcóxialcóxi. A sonda pode ser de cerca de 8 nucleotídeos a cerca de 100 nucleotídeos, ou cerca de 10 a 75, ou cerca de 15 a 50, ou cerca de 20 a 30. Em outro aspecto, as sondas de ácido nucleico da presente invenção são fornecidas em um kit para a identificação das mutações FGFR3 em uma amostra, onde o dito kit compreende um oligonucleotídeo que hibridiza especificamente ou adjacentemente a um local da mutação no ácido nucleico que codifica FGFR3. O kit pode ainda compreender instruções para o tratamento de pacientes com tumores que contêm mutações em FGFR3 com um antagonista de FGFR3 com base no resultado de um teste de hibridização utilizando o kit.
[469] As mutações podem também ser detectadas pela comparação da mobilidade eletroforética de um ácido nucleico amplificado com a mobilidade eletroforética de um ácido nucleico correspondente que codifica o FGFR3 do tipo selvagem. A diferença na mobilidade indica a presença de uma mutação na sequência de ácido nucleico amplificada. A mobilidade eletroforética pode ser determinada por qualquer técnica de separação molecular adequada, por exemplo, em gel de poliacrilamida.
[470] Os ácidos nucleicos podem também ser analisados para detecção de mutações utilizando a Detecção de Mutação Enzimática (EMD) (Del Tito et al., Clinical Chemistry 44:731-739, 1998). A EMD usa a resolvase T4 endonuclease VII de bacteriófago, que faz uma varredura no DNA de fita dupla até detectar esta e clivar distorções estruturais causadas por desajustes no pareamento da dupla fita (mismatches) decorrentes de alterações de ácido nucleico, tal como mutações pontuais, inserções e deleções. A detecção de dois pequenos fragmentos formados por clivagem por resolvase, por exemplo, em eletroforese em gel, indica a presença de uma mutação. Os benefícios do método EMD é o fato ser um protocolo único para identificar mutações pontuais, deleções e inserções analisadas diretamente a partir de reações de amplificação, eliminando a necessidade da purificação de amostras, encurtando o tempo de hibridação, e aumentando a relação sinal-ruído. Amostras mistas contendo um excesso de até 20 vezes o normal de ácidos nucleicos e fragmentos de até 4 kb de tamanho podem ser utilizadas neste teste. No entanto, a varredura por EMD não identifica as mudanças de bases específicas que ocorrem especialmente em amostras positivas para mutação, necessitando, desse modo, de procedimentos de sequenciamento adicionais para identificar a mutação específica, caso necessário. A enzima CEL I pode ser usada da mesma forma que a resolvase T4 endonuclease VII, conforme demonstrado na Patente US 5869245.
[471] Outro kit simples para a detecção de mutações é uma tira teste de hibridização reversa semelhante à hemocromatose StripAssay® (Viennalabs - http://www.bamburghmarrsh.com/pdf/4220.pdf) para a detecção de mutações múltiplas nos genes HFE, TFR2 e FPN1 causando a Hemocromatose. Este ensaio baseia-se na hibridização da sequência específica seguido pela amplificação por PCR. Para os testes de mutação, pode ser aplicado um sistema de detecção baseado em microplacas, enquanto que para os ensaios de mutações múltiplas, um teste de tiras pode ser usado na forma de "macroarranjos". Kits podem incluir reagentes prontos para uso para preparação de amostras, amplificação e detecção da mutação. Protocolos de amplificação Multiplex proporcionam comodidade e permitem a análise de amostras com volumes muito limitados. Usando o formato StripAssay direto, um teste para vinte mutações ou mais pode ser concluído em menos de cinco horas sem a necessidade de equipamentos caros. O DNA é isolado a partir de uma amostra e o ácido nucleico alvo e é amplificado in vitro (por exemplo, por PCR) e marcado com biotina, geralmente em uma única reação de amplificação ("multiplex"). Os produtos da amplificação são então seletivamente hibridizados nas sondas de oligonucleotídeos (tipo selvagem e mutante específico) imobilizados em um suporte sólido como uma tira de teste onde as sondas são imobilizadas como linhas paralelas ou bandas. Os amplicons biotinilados acoplados são detectados através da estreptavidina-fosfatase alcalina e substratos de cor. O referido ensaio pode detectar todo ou qualquer subconjunto de mutações da presente invenção. Com relação a uma banda da sonda mutante específica, um dentre os três padrões de sinalização são possíveis: (i) uma banda apenas para a sonda do tipo selvagem que indica a sequência normal de ácido nucleico, (ii) bandas tanto para o tipo selvagem quanto para a sonda mutante que indica genótipo heterozigoto, e (iii) a banda apenas para a sonda mutante que indica o genótipo mutante homozigoto Assim, em um aspecto, a presente invenção fornece um método para a detecção de mutações da presente invenção que compreende isolar e/ou amplificar uma sequência de ácido nucleico do FGFR3 alvo em uma amostra, de tal forma que o produto da amplificação compreende um ligante, e o produto da amplificação é colocado em contato com uma sonda que compreende um parceiro de ligação detectável ao ligante e a sonda é capaz de hibridizar especificamente a uma mutação descrita na presente invenção, e, em seguida, é detectada a hibridização da referida sonda com o produto de amplificação. Em uma realização, o ligante é biotina e parceiro de ligação compreende a avidina ou estreptavidina. Em uma realização, o parceiro de ligação compreende a esteptoavidina-alcalina, que é detectável com substratos de cor. Em uma realização, as sondas estão imobilizadas, por exemplo, sobre uma tira teste de modo que as sondas complementares para mutações diferentes estão separadas umas das outras. Alternativamente, o ácido nucleico amplificado é marcado com um radioisótopo, caso em que a sonda não precisa conter um marcador detectável.
[472] Alterações de um gene tipo selvagem abrangem todas as formas de mutações, como inserções, inversões, deleções e/ou mutações pontuais. Em uma realização, as mutações são somáticas.
[473] Mutações somáticas são aquelas que ocorrem apenas em certos tecidos, por exemplo, no tecido do tumor, e não são herdadas na linhagem germinal.
[474] As mutações germinativas podem ser encontradas em qualquer tecido do corpo.
[475] A amostra compreendendo por um ácido nucleico alvo pode ser obtida por métodos bem conhecidos no estado da técnica, e que são apropriados para o tipo específico e localização do tumor. A biópsia de tecido é frequentemente usada para se obter uma peça representativa do tecido tumoral. Alternativamente, as células tumorais podem ser obtidas indiretamente na forma de tecidos/fluidos que são conhecidos, ou em que se acredita conter as células tumorais de interesse. Por exemplo, as amostras de lesões de câncer de pulmão podem ser obtidas por ressecção, broncoscopia, aspiração com agulha fina, escovação brônquica, ou a partir de escarro, líquido pleural ou sangue. Os genes mutantes ou produtos gênicos podem ser detectados a partir do tumor ou de outras amostras corpóreas, tais como urina, escarro ou soro. As mesmas técnicas discutidas acima para a detecção de genes alvos mutantes ou produtos gênicos em amostras tumorais podem ser aplicadas a outras amostras corpóreas. As células cancerosas se desprendem a partir dos tumores e aparecem em tais amostras corpóreas. Pelo screening de tais amostras corpóreas, um simples diagnóstico precoce pode ser realizado para esses tipos de câncer. Além disso, o progresso da terapia pode ser monitorado com mais facilidade pela análise de genes alvo ou produtos de genes em tais amostras corpóreas.
[476] Meios para enriquecer uma preparação de tecidos para as células cancerosas são conhecidos no estado da técnica. Por exemplo, o tecido pode ser isolado a partir de cortes de tecidos emblocados em parafina ou a partir de cortes em criostato. As células cancerosas também podem ser separadas das células normais por citometria de fluxo ou captura por microdissecção a laser. Estas, bem como outras técnicas de separação de tumores a partir das células normais, são bem conhecidas no estado da técnica. Se o tecido do tumor é altamente contaminado com células normais, a detecção das mutações pode ser mais difícil, embora técnicas para minimizar a contaminação e/ou falsos positivos/negativos sejam conhecidas, algumas das quais estão descritas abaixo. Por exemplo, uma amostra pode também ser avaliada quanto a presença de um biomarcador (incluindo uma mutação) conhecido que está associado com uma célula tumoral de interesse, mas não com uma célula correspondente normal, ou vice-versa.
[477] A detecção de mutações pontuais nos ácidos nucleicos alvo pode ser obtida por clonagem molecular dos ácidos nucleicos alvo e sequenciamento dos ácidos nucleicos usando métodos bem conhecidos no estado da técnica. Alternativamente, as técnicas de amplificação, como a reação em cadeia da polimerase (PCR) podem ser usadas para amplificar sequências alvo de ácido nucleico diretamente a partir da preparação do DNA genômico do tecido tumoral. A sequência de ácido nucleico das sequências amplificadas pode então ser determinada e as mutações identificadas. As técnicas de amplificação são bem conhecidas no estado da técnica, por exemplo, a reação em cadeia da polimerase conforme descrito em Saiki et al. Science 239:487, 1988; Patentes US 4683203 e US 4683195.
[478] Deve-se notar que o desenvolvimento e a seleção de primers apropriados estão bem estabelecidos no estado da técnica.
[479] A reação em cadeia da ligase, que é conhecido no estado da técnica, também pode ser usada para amplificar sequências alvo de ácido nucleico. Vide, por exemplo, Wu et al. Genomics, vol. 4, pp. 560-569 (1989). Além disso, uma técnica conhecida como PCR alelo-específica também pode ser usada. Vide, por exemplo, Ruano e Kidd, Nucleic Acids Research, vol. 17, págs. 8.392, 1989. Segundo esta técnica, são utilizados primers que hibridizam em suas extremidades 3' a um ácido nucleico alvo de mutação específica. Se a mutação específica não estiver presente, um produto de amplificação não é observado. O sistema de amplificação refratária de mutação (ARMS) também pode ser utilizado, conforme divulgado no Pedido de Patente Europeu EP 0332435, e em Newton et al. Nucleic Acids Research, vol. 17, p.7, 1989. As inserções e deleções de genes também podem ser detectadas através da clonagem, sequenciamento e amplificação. Além disso, sondas de Polimorfismo de Comprimento de Fragmento de Restrição (RFLP) para o gene ou genes em torno marcador podem ser utilizadas para pontuar as alterações de um alelo ou uma inserção em um fragmento polimórfico. O Polimorfismo Conformacional de Fita Simples (SSCP) também pode ser usado para detectar variantes de alterações de base de um alelo. Vide, por exemplo, Orita et al. Proc. Natl. Acad. Sci. USA. 86, págs. 2766-2770, 1989 e Genomics, vol. 5, págs. 874-879, 1989. Outras técnicas para a detecção de inserções e deleções conhecidas no estado da técnica também podem ser usadas.
[480] A alteração de genes do tipo selvagem também pode ser detectada com base na alteração de um produto da expressão de um gene tipo selvagem. Esse produto da expressão inclui mRNA, bem como o produto proteína. As mutações pontuais podem ser detectadas por meio da amplificação e sequenciamento do mRNA ou por meio de clonagem molecular do DNA feita a partir do mRNA. A sequência de cDNA clonada pode ser determinada usando técnicas de sequenciamento de DNA que são bem conhecidas no estado da técnica.
[481] Os pareamentos inadequados são duplex de ácido nucleico hibridizados que não são 100% complementares. A falta de complementaridade total pode ser devido a deleções, inserções, inversões, substituições ou mutações que alteram o quadro de leitura (mutações frameshift). A detecção de pareamentos inadequados pode ser usada para detectar mutações pontuais em um ácido nucleico alvo. Embora estas técnicas possam ser menos sensíveis do que sequenciamento, elas são mais simples de serem executadas em um grande número de amostras de tecido. Um exemplo de uma técnica de clivagem de pareamentos inadequado é o método de proteção de RNase, que é descrito em detalhes em Winter et al. Proc. Natl. Acad. Sci. USA., vol. 82, pág. 7575, 1985 e Meyers et al. Science, vol. 230, p. 1242, 1985. Por exemplo, um método da presente invenção pode envolver o uso de uma ribosonda marcada que é complementar aos ácidos nucleicos alvo humano tipo selvagem. O ribosonda e o ácido nucleico alvo derivado da amostra de tecido são anelados (hibridizados) juntos e posteriormente digeridos com a enzima RNase A, que é capaz de detectar alguns pareamentos inadequados na estrutura duplex de RNA. Se for detectada um pareamento inadequado pela RNase A, esta cliva no sítio do pareamento inadequado. Desse modo, quando a preparação de RNA anelada é separada em um gel por eletroforese, se um pareamento inadequado foi detectado e clivado pela RNase A, um produto de RNA menor será observado quando comparado com o RNA duplex de comprimento total para a ribosonda e o mRNA ou DNA. A ribosonda não precisa ser de comprimento total do mRNA ou gene alvo, mas pode conter uma parte do ácido nucleico alvo, que abrange a posição suspeita de mutação. Se a ribosonda compreende apenas um segmento de ácido nucleico do mRNA ou gene alvo, pode ser desejável usar várias destas sondas para detectar toda a sequência de ácido nucleico alvo que possui pareamento inadequado, se desejado.
[482] De maneira similar, as sondas de DNA podem ser usadas para detectar pareamentos inadequados, por exemplo, através da clivagem enzimática ou química. Vide por exemplo, Cotton et al., Proc. Natl. Acad. Sci. USA., vol. 85, 4397, 1988 e Shenk et al. Proc. Natl. Acad. Sci. USA., vol. 72, p. 989, de 1975. Alternativamente, pareamentos inadequados podem ser detectados por alterações na mobilidade eletroforética de dúplex pareados inadequadamente em relação aos dúplex pareados corretamente. Vide, por exemplo, Cariello, Human Genetics, vol. 42, p. 726, de 1988. Com qualquer ribosonda ou sondas de DNA, o ácido nucleico do mRNA ou DNA alvo que podem conter uma mutação pode ser amplificado antes da hibridação. As alterações no ácido nucleico do DNA alvo também podem ser detectadas usando a hibridização Southern, especialmente se as mudanças são rearranjos grosseiros, tais como deleções e inserções.
[483] As sequências de ácido nucleico do DNA alvo que foram amplificadas também podem ser selecionadas utilizando sondas alelo- específicas. Estas sondas são oligômeros de ácido nucleico, cada qual contém uma região de ácido nucleico do gene alvo portadora de uma mutação conhecida. Por exemplo, um oligômero pode ser de cerca de 30 nucleotídeos de comprimento, correspondente a uma parte da sequência do gene alvo. Pelo uso de uma bateria de tais sondas alelo-específicas, os produtos da amplificação de ácidos nucleicos alvos podem ser rastreados para identificar a presença de uma mutação previamente identificadas no gene alvo. A Hibridização de sondas alelo- específica das sequências de ácido nucleico alvo amplificadas pode ser executada, por exemplo, em um filtro de nylon. A hibridização de uma sonda específica, sob condições estringentes de hibridização indica a presença da mesma mutação no tecido tumoral de acordo com a sonda alelo-específica.
[484] A alteração de genes alvo do tipo selvagem também pode ser detectada pelo rastreio de alteração da proteína tipo selvagem correspondente. Por exemplo, anticorpos monoclonais imunorreativos com um produto do gene alvo podem ser usados para o rastreamento no tecido, por exemplo, um anticorpo que se liga a uma posição mutada específica (proteína). Por exemplo, um anticorpo que é usado pode ser um que se ligue a um éxon excluído (por exemplo, éxon 14), ou que se ligue a um epítopo conformacional compreendendo uma parcela excluída da proteína alvo. A ausência do antígeno cognato indica uma mutação. Anticorpos específicos para os produtos dos alelos mutantes também poderiam ser usados para detectar produto do gene mutante. Os anticorpos podem ser identificados a partir de bibliotecas de exibição por fagos. Tais testes imunológicos podem ser feitos em qualquer formato conveniente conhecido no estado da técnica. Estes incluem Western blot, imuno- histoquímica e ensaios ELISA. Todos os meios para a detecção de uma proteína alterada podem ser usados para detectar a alteração dos genes alvo tipo selvagem.
[485] Os pares de primers são úteis para a determinação da sequência de nucleotídeos de um ácido nucleico alvo usando técnicas de amplificação de ácido nucleico, tal como a reação em cadeia da polimerase. Os pares de primers de DNA de fita simples podem ser anelados às sequências dentro ou flanqueando a sequência de ácido nucleico alvo para a amplificação da sequência alvo. Primers alelo-específico também podem ser usados. Tais primers anelam apenas com a sequência alvo mutante e, portanto, só amplificam um produto há a presença da sequência alvo mutante como um molde. A fim de facilitar a posterior clonagem das sequências amplificadas, os primers podem possuir sequências com sítios para a ação de enzimas de restrição em suas extremidades. Tais sítios e enzimas são bem conhecidos no estado da técnica. Os primers podem ser sintetizados através de técnicas que são bem conhecidas no estado da técnica. De modo geral, os primers podem ser produzidos por máquinas de síntese de oligonucleotídeos que são disponíveis comercialmente. O desenvolvimento de primers específicos é bem conhecido no estado da técnica.
[486] Sondas de ácidos nucleicos são úteis para uma série de propósitos. Elas podem ser usadas na hibridação Southern do DNA genômico e no método de proteção por RNase para a detecção de mutações pontuais já discutidas acima. As sondas podem ser usadas para detectar produtos de amplificação de ácidos nucleicos alvos. Elas também podem ser usadas para detectar pareamentos inadequados com o gene ou mRNA do tipo selvagem usando outras técnicas. Pareamentos inadequados podem ser detectados usando enzimas (por exemplo, Nuclease S1), produtos químicos (por exemplo, hidroxilamina ou tetróxido de ósmio e piperidina), ou alterações na mobilidade eletroforética de híbridos pareados inadequadamente em comparação com híbridos totalmente pareados. Estas técnicas são conhecidas no estado da técnica. Vide, Novack et al., Proc. Natl. Acad. Sci. USA., vol. 83, p. 586, de 1986. De modo geral, as sondas são complementares às sequências fora do domínio quinase. Uma bateria inteira de sondas de ácidos nucleicos pode ser utilizada para compor um kit para detecção de mutações nos ácidos nucleicos alvos. O kit permite a hibridização de uma grande região de uma sequência alvo de interesse. As sondas podem sobrepor-se umas com as outras ou podem ser contíguas.
[487] Se uma ribosonda é usada para detectar pareamentos inadequados com o mRNA, ela é geralmente complementar ao mRNA do gene alvo. A ribosonda, portanto, é uma sonda antisense de modo que ela não codifica o produto gênico correspondente, pois é complementar à fita sense. A ribosonda em geral será marcada com um material radioativo, colorimétrico, ou fluorimétrico, que pode ser feito por qualquer método conhecido no estado da técnica. Se a ribosonda é usada para detectar pareamentos inadequados com o DNA, ela pode ser de qualquer polaridade, ou seja, sense ou antisense. Da mesma forma, as sondas de DNA também podem ser usadas para detectar pareamentos inadequados.
[488] Em alguns casos, o câncer superexpressa ou não o FGFR3. A superexpressão do receptor pode ser determinada em um ensaio diagnóstico ou prognóstico, avaliando os níveis aumentados da proteína receptor presente na superfície de uma célula (por exemplo, por meio de um ensaio de imunohistoquímica; IHQ). Alternativamente, ou adicionalmente, podem ser mensurados os níveis de ácido nucleico que codificam o receptor na célula, por exemplo, por meio da hibridização in situ fluorescente (FISH; vide, publicação WO 98/45479 publicado em outubro de 1998), técnicas de Southern blotting, ou reação em cadeia da polimerase (PCR), como PCR quantitativo em tempo real (qRT-PCR). Com exceção dos ensaios acima, vários ensaios in vivo e ex vivo estão disponíveis aos profissionais hábeis no assunto. Por exemplo, um ensaio que possa expor célula do corpo do paciente a um anticorpo que é opcionalmente marcado com um marcador detectável, por exemplo, um isótopo radioativo, e a ligação do anticorpo com as células do paciente pode ser avaliada, por exemplo, por varredura externa da radioatividade ou pela análise de uma biópsia retirada de um paciente que foi exposto previamente ao anticorpo.
[489] A terapia combinada da presente invenção pode ainda compreender um ou mais agente(s) quimioteráopico(s). A administração combinada inclui a coadministração ou a administração concomitante, usando formulações distintas ou uma única formulação farmacêutica, e a administração consecutiva em qualquer ordem, de preferência onde haja um período de tempo, em que ambos (ou todos) os agentes exerçam simultaneamente suas atividades biológicas.
[490] O agente quimioterápico, se administrado, é geralmente administrado nas dosagens conhecidas destes, ou, opcionalmente em dosagens reduzidas devido à ação combinada das drogas ou efeitos colaterais negativos atribuídas à administração do agente quimioterápico antimetabólito. A preparação e esquemas de dosagem desses agentes quimioterápicos podem ser utilizados de acordo com as instruções dos fabricantes, ou conforme determinado empiricamente pelo médico qualificado.
[491] Vários agentes quimioterápicos que podem ser combinados são divulgados no presente.
[492] Em alguns exemplos de realização, os agentes quimioterápicos a serem combinados são selecionados a partir grupo que consiste em lenalidomida (REVLIMID), inibidores de proteossoma (como o bortezomib (Velcade) e PS342), toxóides da bora (inclusive docetaxel e paclitaxel), vinca (como vinorelbina ou vinblastina), composto de platina (tais como a carboplatina ou cisplatina), inibidores da aromatase (tais como o letrozol, anastrazol, ou exemestano), anti-estrógenos (por exemplo, fulvestrant ou tamoxifeno), etoposide, tiotepa, ciclofosfamida, pemetrexato, metotrexato, doxorrubicina lipossomal, doxorrubicina lipossomal peguilada, capecitabina, gemcitabina, meltalina, doxorubicina, vincristina, inibidor da COX-2 (por exemplo, celecoxib), ou esteróides (por exemplo, dexametasona e prednisona). Em alguns exemplos de realização por exemplo, realizações envolvendo tratamento de mieloma múltiplo t(4;14)+, a dexametasona e lenalidomida, ou dexametasona, ou bortezomib, ou vincristina, doxorrubicina e dexametasona, ou talidomida e dexametasona, ou doxorrubicina lipossomal, vincristina e dexametasona, ou lenalidomida e dexametasona, ou bortezomib e dexametasona, ou bortezomib, doxorrubicina e dexametasona são combinados.
[493] Em alguns exemplos de realização (por exemplo, realizações envolvendo o câncer de bexiga), são combinados: gemcitabina e cisplatina, ou um taxano (por exemplo, paclitaxel, docetaxel), ou pemetrexato, ou metotrexato, vinblastina, doxorrubicina e cisplatina, ou carboplatina, ou mitomicina C em combinação com 5-fluorouracila, ou cisplatina, ou cisplatina e 5-fluorouracila.
[494] Os agentes terapêuticos utilizados na presente invenção serão formulados, dosados e administrados de forma coerente com a boa prática clínica. Fatores que devem ser considerados neste contexto incluem o distúrbio específico a ser tratado, o indivíduo específico a ser tratado, a condição clínica do paciente, a causa do distúrbio, o local de entrega do agente, o método de administração, a programação da administração, a interação droga-droga dos agentes a serem combinados, e outros fatores conhecidos pelos médicos.
[495] As formulações terapêuticas são preparadas usando métodos padrões conhecidos no estado da técnica pela mistura do ingrediente ativo possuindo o grau desejado de pureza com opcionais veículos fisiologicamente aceitáveis, excipientes ou estabilizantes (Remington's Pharmaceutical Sciences (edição 20), ed. A. Gennaro, 2000, Lippincott, Wilkins Williams &, Philadelphia, PA). Veículos aceitáveis incluem; soro fisiológico ou tampões como o fosfato, citrato e outros ácidos orgânicos, incluindo antioxidantes, ácido ascórbico, polipeptídeos de baixo peso molecular (inferior a cerca de 10 resíduos), proteínas, tal como albumina sérica, gelatina ou imunoglobulinas; polímeros hidrofílicos, tais como polivinilpirrolidona, aminoácidos como a glicina, glutamina, asparagina, arginina ou lisina; monossacarídeos, dissacarídeos e outros carboidratos, incluindo glicose, manose ou dextrina; agentes quelantes tal como o EDTA, alcoóis de açucares tais como sacarose, manitol, trealose ou sorbitol; contra-íons formadores de sal como o sódio; tensoativos não-iônicos tais como TWEEN®, PLURONICS® ou polietileno glicol (PEG).
[496] Opcionalmente, mas preferencialmente, a formulação contém um sal farmaceuticamente aceitável, preferivelmente cloreto de sódio, e de preferência em concentrações próximas das concentrações fisiológicas. Opcionalmente, as formulações da presente invenção podem conter um conservante farmaceuticamente aceitável. Em algumas realizações a concentração de conservante varia de 0,1 a 2,0%, tipicamente v/v. Conservantes adequados incluem aqueles conhecidos na técnica farmacêutica. Álcool benzílico, fenol, m-cresol, metilparabeno e propilparabeno são conservantes preferenciais. Opcionalmente, as formulações da presente invenção podem incluir um tensoativo farmaceuticamente aceitável em uma concentração de 0,005 a 0,02%.
[497] A presente formulação também pode conter mais de um composto ativo de acordo com a necessidade para a indicação específica sendo tratada, preferencialmente compostos ativos com atividades complementares que não sejam prejudiciais entre si. Tais moléculas estão adequadamente presentes na combinação em quantidades que são eficazes para o propósito pretendido.
[498] Os ingredientes ativos podem também ser armazenados em microcápsula preparadas, por exemplo, por técnicas de coacervação ou por meio de polimerização interfacial, por exemplo, hidroximetilcelulose ou microcápsula de gelatina e microcápsula de poli-(metilmetacrilato), respectivamente, em sistemas de fornecimento de drogas coloidais (por exemplo, lipossomas, microesferas de albumina, microemulsões, nano-partículas e nanocápsulas) ou em macroemulsões. Tais técnicas são divulgadas em Remington’s Pharmaceutical Sciences, supra.
[499] Podem ser fabricadas preparações de liberação prolongada. Exemplos apropriados de preparações de liberação prolongada incluem matrizes semipermeáveis de polímeros hidrofóbicos sólidos que contenham o anticorpo, que se encontram na forma de artigos moldados, tais como filmes ou microcápsulas. Exemplos de matrizes de liberação prolongada incluem poliésteres, hidrogéis (tais como poli-(metacrilato de 2-hidroxietila) ou álcool poli- (vinílico)), poli-lactídeos (patente US 3.773.919), copolímeros de ácido L- glutâmico e L-glutamato de g etila, acetato de etilenovinila não-degradável, copolímeros degradáveis de ácido láctico e ácido glicólico, tal como LUPRON DEPOT™ (microesferas injetáveis compostas de copolímero de ácido láctico e ácido glicólico e acetato de leuprolide) e ácido poli-D-(-)-3-hidroxibutírico. Embora polímeros tais como etilenovinilacetato e ácido láctico-ácido glicólico permitam a liberação de moléculas por mais de 100 dias, certos hidrogéis liberam proteínas por períodos de tempo mais curtos. Quando encapsulados, os anticorpos permanecem no corpo por um longo período, eles podem desnaturar ou se agregarem como resultado da exposição à umidade a 37 °C, resultando na perda da atividade biológica e possíveis mudanças na imunogenicidade. Estratégias racionais podem ser concebidas para a estabilização, dependendo do mecanismo envolvido. Por exemplo, se é revelado que mecanismo de agregação é a formação de pontes S-S intermoleculares através do intercâmbio tio-dissulfeto, a estabilização pode ser obtida pela modificação de resíduos sufidrila, liofilização de soluções ácidas, controlando o conteúdo de umidade, utilizando aditivos adequados e desenvolvendo composições de matriz polimérica específicas.
[500] Os agentes terapêuticos da presente invenção são administrados a um paciente humano, de acordo com métodos conhecidos, como a administração intravenosa em bolus ou por infusão contínua durante um período de tempo, por via intramuscular, intraperitoneal, intracerobrospinal, subcutânea, intra-articular, intra-sinovial, intratecal, via oral, tópica, ou por inalação. Uma estratégia ex vivo também pode ser usada para aplicações terapêuticas. Estratégias ex vivo envolvem a transfecção ou transdução de células obtidas a partir do indivíduo com um polinucleotídeo que codifica uma antagonista FGFR3. As células transfectadas ou transduzidas são então devolvidas ao indivíduo. As células podem ser qualquer uma dentre uma grande variedade de tipos celulares, incluindo, sem se limitar, a células hematopoiéticas (por exemplo, células da medula óssea, macrófagos, monócitos, células dendríticas, células T ou células B), fibroblastos, células epiteliais, células endoteliais, queratinócitos ou células musculares.
[501] Por exemplo, se o antagonista de FGFR3 é um anticorpo, o anticorpo é administrado por qualquer via adequada, inclusive a via parenteral, subcutânea, intraperitoneal, intrapulmonar e intranasal, e, se for desejado um tratamento imunossupressor local, a administração intralesional. As infusões parenterais incluem administração intramuscular, intravenosa, intra-arterial, intraperitoneal ou subcutânea. Além disso, o anticorpo é administrado adequadamente por meio de infusão pulsada, particularmente com doses decrescentes do anticorpo. Preferencialmente a dosagem é dada por injeções, mais preferencialmente por injeções intravenosas ou subcutâneas, dependendo, em parte, se a administração é breve ou crônica.
[502] Em outro exemplo, o composto antagonista de FGFR3 é administrado localmente, por exemplo, por injeção direta, quando o distúrbio ou a localização do tumor o permita, e as injeções podem ser repetidas periodicamente. O antagonista de FGFR3 também pode ser entregue sistemicamente para o indivíduo ou pode ser entregue diretamente para as células tumorais, por exemplo, para a um tumor ou leito tumoral após a excisão cirúrgica do tumor a fim de prevenir ou reduzir a recidiva local ou metástase.
[503] A administração de agentes terapêuticos em combinação é geralmente realizada durante um período de tempo definido (normalmente minutos, horas, dias ou semanas, dependendo da combinação selecionada). A terapia combinada pretende abranger a administração destes agentes terapêuticos de forma sequencial, ou seja, onde cada agente terapêutico é administrado em um momento diferente, assim como a administração destes agentes terapêuticos, ou pelo menos dois dos agentes terapêuticos, de uma forma substancialmente simultânea.
[504] O agente terapêutico pode ser administrado pela mesma via ou por vias diferentes. Por exemplo, o anticorpo anti-FGFR3 na combinação pode ser administrado por injeção intravenosa, enquanto um agente quimioterápico da combinação pode ser administrado por via oral. Alternativamente, por exemplo, ambos os agentes terapêuticos podem ser administrados por via oral, ou ambos os agentes terapêuticos podem ser administrados por injeção intravenosa, dependendo dos agentes terapêuticos específicos. A sequência no qual os agentes terapêuticos são administrados também varia de acordo com os agentes específicos.
[505] Dependendo do tipo e da severidade da doença, cerca de 1 μg/kg a 100 mg/kg de cada agente terapêutico é uma possível dose candidata inicial para administração ao paciente, seja, por exemplo, por meio de uma ou mais administrações separadas ou por meio de infusão contínua. Uma dosagem diária típica poderá variar em cerca de 1 μg/kg a cerca de 100 mg/kg ou mais, dependendo dos fatores mencionados acima. Para administrações repetidas ao longo de vários dias ou mais, dependendo da condição, o tratamento é mantido até que o câncer seja tratado, conforme mensurado pelos métodos descritos acima. Entretanto, outros regimes de dosagens podem ser úteis.
[506] O presente pedido contempla a administração do anticorpo para FGFR3 por terapia gênica. Vide, por exemplo, a publicação WO96/07321 publicado em 14 de março de 1996 que se refere à utilização da terapia genética para gerar anticorpos intracelulares.
[507] Em outro aspecto da presente invenção, é fornecido um artigo manufaturado que contém materiais úteis para o tratamento, prevenção e/ou diagnóstico dos distúrbios descritos anteriormente. O artigo manufaturado compreende um recipiente e um rótulo ou bula sobre o recipiente ou associado a ele. Recipientes apropriados incluem, por exemplo, garrafas, ampolas, seringas etc. Os recipientes podem ser formados a partir de uma série de materiais tais como vidro ou plástico. O recipiente guarda uma composição que é por si só, ou em combinação com outra(s) composição(ões), eficaz para o tratamento, prevenção e/ou diagnóstico da condição e pode ter uma porta de acesso estéril (por exemplo, o recipiente pode ser uma bolsa de solução intravenosa ou uma ampola que contém uma tampa perfurável por uma agulha de injeção hipodérmica). Pelo menos um agente ativo na composição é um anticorpo da presente invenção. O rótulo ou bula indica que a composição é utilizada para o tratamento da condição de escolha, como o câncer. Além disso, o artigo manufaturado pode compreender (a) um primeiro recipiente com uma composição ali contida, em que a composição compreende um anticorpo da presente invenção, e (b) um segundo recipiente com uma segunda composição ali contida, em que a segunda composição compreende um agente citotóxico adicional. O artigo de manufatura neste exemplo de realização pode ainda compreender uma bula indicando que a primeiro e segunda composição de anticorpos podem ser usadas para tratar uma condição específica, por exemplo, o câncer. Alternativamente ou adicionalmente, o artigo manufaturado pode compreender ainda um segundo (ou terceiro) recipiente que compreende um tampão farmaceuticamente aceitável, tal como água bacteriostática para injeção (BWFI), solução salina tamponada com fosfato, solução de Ringer e solução de dextrose. Ele pode incluir adicionalmente outros materiais desejáveis do ponto de vista comercial e do usuário, incluindo outros tampões, diluentes, filtros, agulhas e seringas.
[508] Os seguintes são exemplos de métodos e composições da presente invenção. Entende-se que diversas outras realizações podem ser praticadas, dada a descrição geral fornecida acima.
[509] A linhagem de células RT4 foi obtida a partir da Coleção Americana de Cultivo de Tipo (American Type Culture Collection - ATCC). As linhagens de células RT112, OPM2 e Ba/F3 foram adquiridos da coleção alemão de Microrganismos e Culturas de Células (DSMZ, (Alemanha)). A linhagem de células de mieloma múltiplo KMS11 foi gentilmente cedida pelo Dr. Takemi Otsuki de Kawasaki Medical School (Japão). A linhagem de células de câncer de bexiga TCC- 97-7 foi gentilmente cedida pelo Dr. Margaret Knowles do Hospital Universitário de St. James (Leeds, UK). A linhagem de células UMUC-14 foi obtida do Dr. H.B. Grossman (atualmente na Universidade do Texas- MD Anderson Cancer Center, TX). As células foram mantidas em meio RPMI suplementado com 10% de soro fetal bovino (FBS) (Sigma), 100 U/ml de penicilina, 0,1 mg/ml de estreptomicina e L-glutamina em condições de 5% de CO2 a 37 °C.
[510] Células UMUC -14 foram cultivadas em meio sem cisteína, tratadas com R3Mab ou DTNB por 3 hr, e os lisados celulares foram submetidos a análise por immunoblotting sob condições redutoras ou não redutoras. Para os estudos de dimerização in vitro, o FGFR3-IIIbS249C (resíduos 143-374) foi clonado no vetor pAcGP67A e expresso em células T.ni Pro. A proteína recombinante foi purificada através de uma coluna Ni-NTA seguido epla purificação em coluna Superdex S200. O FGFR3S249C dimérico foi eluído em 25 mM de Tris (pH 7,5) e 300 mM de NaCl. O R3Mab (1 mM) foi incubado com o dímero FGFR3S249C (0,1 mM) a 37 ° C sob as seguintes condições: 100 mM KH2PO4 (pH 7,5), 25 μM de DTT, 1 mM de EDTA e 0,75 mg/ml de BSA. As alíquotas da reação foram tomadas em momentos indicados e a reação foi interrompida pela adição de tampão de amostra sem β-mercaptoetanol. O dímero-monômero foi analisado por immunoblott.
[511] Todos os estudos foram aprovados pelo Comitê Institucional de Uso e Cuidados Animais da Genentech. Camundongos fêmeas nu/nu ou camundongos CB17 com imunodeficiência severa combinada (SCID) de 6 a 8 semanas de idade foram adquiridos da Charles River Laboratory (Hollister, CA). Camundongos nude fêmeas atímicos foram obtidos do Instituto Nacional de Câncer (National Cancer Institute-Frederick Cancer Center). Os camundongos foram mantidos sob condições específicas livres de patógenos. Células RT112 estáveis com shRNA (7 x106), RT112 (7 x106), Ba/F3-FGFR3S249C (5 x106), OPM2 (15 x106), ou células KMS11 (20 x106) foram implantadassubcutaneamente no flanco de camundongos em um volume de 0,2 ml em HBSS/Matrigel (1:1 v/v, BD Biosciences). As células UMUC -14 (5 x106) foram implantados sem matrigel. Os tumores foram mensurados duas vezes por semana usando um compasso de calibre, e o volume tumoral foi calculado usando a fórmula: V=0,5 a x b2, onde a e b são o comprimento e a largura do tumor, respectivamente. Quando o volume tumoral médio atingiu 150 a 200 mm3, os ratos foram randomizados em grupos de 10 e foram tratados duas vezes por semana com injeção intraperitoneal (ip) de R3Mab (0,3-50 mg/kg), ou IgG1 controle humano diluído em HBSS. Os animais do grupo controle receberam apenas veículo (HBSS).
[512] Os dados agrupados são expressos como média +/- EPM. Os testes t de Student não pareados (bicaudal) foram utilizados para comparação entre dois grupos. Um valor de P < 0,05 foi consideradoestatisticamente significativo em todos os experimentos. GERAÇÃO DE CÉLULAS ESTÁVEIS FGFR3 SHRNA
[513] Três FGFR3 shRNA independentes foram clonados em um vetor pHUSH conforme descrito (1). A sequência utilizada dos shRNAs para FGFR3 nos estudos foram as seguintes:shRNA2:5’GATCCCCGCATCAAGCTGCGGCATCATTCAAGAGATGATGC CGCAGCTTGATGCTTTTTTGGAAA (SEQ ID NO:192);shRNA4:5’-GATCCCCTGCACAACCTCGACTACTATTCAAGAGATAGTAGT CGAGGTTGTGCATTTTTTGGAAA-3’ (SEQ ID NO:193);shRNA6: 5’-GATCCCCAACCTCGACTACTACAAGATTCAAGAGATCTTGTA GTAGTCGAGGTTTTTTTTGGAAA-3’ (SEQ ID NO:194).[001] Todas as construções foram confirmadas por sequenciamento. O shRNA controle EGFP foi descrito em nosso estudo anterior (50). O retrovírus contendo shRNA foi produzido por cotransfecção de células embaladas GP2-293 (Clontech Laboratories, Mountain View, CA) com VSV -G (Clontech Laboratories) e as construções de shRNA pHUSH-FGFR3, e os sobrenadantes virais foram coletados 72 horas após a transfecção, e os detritos celulares foram retirados por centrifugação para o experimento de transdução.
[514] As células RT112 foram mantidas em meio RPMI 1640 contendo FBS sem tetraciclina (Clontech Laboratories), e transduzidas com o sobrenadante retroviral na presença de 4 mg/ml polibreno. 72 horas após a infecção, 2 mg/ml de puromicina (Clontech Laboratories) foi adicionada ao meio para selecionar clones estáveis expressando o shRNA. As células estáveis foram isoladas, tratadas com 0,1 ou 1 μg/ml doxiciclina (Clontech Laboratories) durante 4 dias, e o silenciamento gênico (knockdown) induzível da expressão da proteína FGFR3 foi avaliada pela técnica de Western blotting. As análises do ciclo celular foram realizadas conforme descrito (51).SELEÇÃO DE ANTICORPOS FAGO ESPECÍFICOS PARA FGFR3
[515] Bibliotecas de anticorpo de fago humano com diversidades sintéticas nas regiões determinantes de complementaridade selecionadas (H1, H2, H3, L3), mimetizando a diversidade natural do repertório de IgG humano, foram utilizadas para seleção. Fragmentos Fab foram exibidos de forma bivalente na superfície de partículas de bacteriófago M13 (52). IgD2-D3 com His- marcada de FGFR3-IIIb e IIIc humano foram usados como antígenos. Placas de 96 poços Maxisorp Immunoplates (Nunc) foram revestidas durante a noite a 4 °C com a proteína FGFR3-IIIb-His ou proteína FGFR3-IIIC-His (10 μg/ml) e bloqueados por uma hora com tampão PBST (PBS com 0,05% de Tween-20) suplementado com BSA 1%. As bibliotecas de fago anticorpo foram adicionadas e incubadas durante a noite a temperatura ambiente (RT). As placas foram lavadas com tampão PBST e os fagos ligados foram eluidos com HCl 50 mM e NaCl 500 mM por 30 minutos e neutralizados com igual volume de 1M de base Tris. Os fagos recuperados foram amplificados em células E. coli XL-1 blue. Durante as etapas de seleção subsequentes, o tempo de incubação dos anticorpos fago foi reduzido para 2 horas e a estringência das lavagens da placa foi gradualmente aumentada (53). Anticorpos de fago únicos e específicos que se ligaram as isoformas IIIb e IIIc do FGFR3 foram identificados por fago-ELISA e sequenciamento de DNA. Dos 400 clones triados, quatro foram selecionados para reformatar as IgGs de comprimento total pela clonagem das regiões VL e VH de clones individuais em vetores LPG3 e LPG4, respectivamente, transitoriamente expressos em células de mamíferos, e purificadas com colunas de proteína A (54). O clone 184,6 foi selecionado para a maturação por afinidade.
[516] Para a maturação de afinidade, um fagomídeo exibindo Fab monovalente na superfície do bacteriófago M13 (52) serviu como modelo de biblioteca para a realização do enxerto dos domínios variáveis de cadeia leve (VL) e cadeia pesada (VH) do Ab fago. Os códons de parada foram então incorporados na CDR-L3. Uma estratégia de randomização leve foi utilizada para maturação por afinidade conforme descrito (53). Duas combinações diferentes de alças CDR, H1/H2/L3, H3/L3 ou L1/L2/H3 foram selecionadas para arandomização. Para seleção de clones maturados por afinidade, bibliotecas de fago foram classificadas contra proteína FGFR3-IIIb ou IIIc-His, submetidas a seleção para um primeiro ciclo, seguido por quatro ciclos de seleção em fase de solução conforme descrito (52). Após cinco rodadas de seleção, um fago-ELISA de alto rendimento competitivo de um único ponto foi utilizado para triar rapidamente clones com alta afinidade conforme descrito (55). Clones com baixa relação de absorbância a 450 nm na presença de 10 nM de FGFR3-His, na ausência de FGFR3-His foram escolhidos para uma melhor caracterização.
[517] Os clones 184.6.1, 184.6.21, 184.6.49, 184.6.51, 184.6.58, 184.6.62 e 184.6.92 reduziram significativamente a viabilidade das linhagens de células Ba/F3-FGFR3-IIIb, Ba/F3-FGFR3-IIIc e Ba/F3- FGFR3-S249C e o clone 184.6.52 reduziu significativamente a viabilidade linhagem celular Ba/F3- FGFR3-S249C. O aumento da atividade inibitória variou de cerca de 50 vezes (clone 184.6.52) a cerca de 100 vezes (clones 184.6.1, 184.6.21, 184.6.49, 184.6.51, 184.6.58, 184.6.62 e 184.6.92) acima do clone parental 184,6,dependendo da linhagem de células analisadas. A cinética de ligação dos clones 184.6.1, 184.6.58, e 184.6.62 para o FGFR3-IIIb e FGFR3-IIIc foi determinada utilizando o ensaio BIAcore da seguinte forma:
[518] Os clones 184.6.1, 184.6.58 e 184.6.62 também exibiram inibição melhorada da sinalização a jusante de FGFR3 nas linhagens de células Ba/F3-FGFR3, RT112 e OPM2.
[519] O clone 184.6.1 foi selecionado. Uma modificação de sequência, N54S, foi introduzida no HVR H2 no resíduo 54, para melhorar a fabricação, criando o clone 184.6.1N54S. Os clones 184.6.1 e 184.6.1N54S exibiram cinéticas de ligação comparáveis (mensuradas em ensaios Biacore) e atividade comparável no ensaio de viabilidade celular com células Ba/F3. Variantes HVR H2 adicionais foram gerados: N54S foi introduzido no clone 184.6.58, e N54G, N54A, ou N54Q foram introduzidas no clone 184.6.1 e 184.6.58. Esses clones apresentaram atividade comparável no ensaio de viabilidade celular para clones Ba/F3 parentais 184.6.1 ou 184.6.58.
[520] Outra modificação de sequência, a D30E, foi introduzida na HVR L1 do clone 184.6.1N54S, criando o clone 184.6.1NSD30E. O clone 184.6.1N54S e clone 184.6.1NSD30E exibiram uma cinética de ligação comparável e atividade comparável no ensaio de viabilidade celular com células BA/F3 aos clones parentais 184.6.1 ou 184.6.58.
[521] Conforme usado no presente, “Mab R3” refere-se a anticorpos anti-FGFR3 clones 184.6.1N54S, 184.6.1, ou 184,6. O clone184.6.1N54S foi usado nas figuras e experimentos referenciados “R3Mab”, exceto nos experimentos com os resultados mostrados nas seguintes figuras (para as quais o anticorpo utilizado é exibido em parênteses): Figuras 9B (clone 184.6.1), 10 (clone 184.6), 11A e B (clone 184.6), 13 (clone 184.6.1), 14A (clone 184.6.1), 14B, G, e H (clone 184.6), 19 (clone184.6.1), e 22B e C (clone 184.6.1).ANÁLISE POR BIACORE/RESSONÂNCIA PLASMÔNICA DE SUPERFÍCIE (SRP) PARA DETERMINAR AS AFINIDADES DE LIGAÇÕES DE ANTICORPOS
[522] As afinidades de ligação de R3Mab para o FGFR3 foram mensuradas por BIAcore / ressonância plasmônica de superfície (SRP) utilizando um instrumento BIAcore™-3000 conforme descrito (52) com as seguintes modificações. O R3Mab foi revestido diretamente no chip biosensor CM5 para alcançar aproximadamente 400 unidades de resposta (RU). Para as medições cinéticas, diluições em série de duas vezes de proteína FGFR3-IIIa ou IIIc-His (iniciando a partir de 67 nM) foram injetadas em tampão PBST a 25°C a uma velocidade de fluxo de 30 μl/minuto. A velocidade de associação (kon por mol/s) e a velocidade de dissociação (koff por s) foram calculadas utilizando um modelo de ligação simples um-para-um de Langmuir (BIAcore Evaluation Software version 3.2). A constante do equilíbrio de dissociação (Kd - por mol) foi calculada como a relação koff/kon.
[523] As afinidades de ligação de anticorpos do hibridoma de camundongo para FGFR3 foram mensuradas por Biacore/SRP da seguinte forma. O FGFR3-IIIb e IIIc humano foi acoplado em três células de fluxo (FC) diferentes, FC2, FC3 e FC4, de um chip sensor CM5 BIACORETM para alcançar uma unidade de resposta (RU) de cerca de 50 RU. A imobilização foi realizada por meio de grupos aminos através de acoplamentos aleatórios usando um protocolo fornecido pelo fabricante. Sensogramas foram registrados quanto a ligação de anti-FGFR3 de IgG murino derivado de hibridoma ou o fragmento Fab a essas superfícies a 25 °C pela injeção de uma série de soluções que vão desde a 250 nm a 0.48 nM em incrementos de 2 vezes a uma velocidade de fluxo de 30 μl/min. Entre cada injeção, 10mM de Glicina-HCl pH 1,7 foi utilizado como tampão para regenerar o chip sensor. O sinal da célula de referência (FC1) foi subtraído a partir do sensograma observado na FC2, FC3 e FC4. As constantes cinéticas foram calculadas pelo ajuste de regressão não linear dos dados de acordo com um modelo de Langmuir de ligação 1:1 usando o software de avaliação do BIAcore (versão 3.2) fornecido pelo fabricante.ESTUDOS DE LIGAÇÃO POR ELISA
[524] cDNAs codificando os domínios extracelulares (ECD) do FGFR1-IIIb, IIIc, FGFR2, IIIb e IIIc, FGFR3 - IIIb e IIIc e FGFR4 humanos foram clonados em um vetor baseado no pRK para gerar proteínas quiméricas FGFR- Fc humano. As proteínas recombinantes foram produzidas pela transfecção transitória em células de ovário de hamster chinês (CHO) e purificadas através de cromatografia de afinidade em proteína A. Para testar a ligação dos anticorpos aos FGFRs humanos, placas de 96 poços Maxisorp (Nunc) foram revestidas durante a noite a 4 °C com 50 μl de 2 μg/ml de proteínas quiméricas FGFR ECD-Fc humano. Após o bloqueio com salina tamponada com fosfato (PBS)/BSA 3%, o anticorpo FGFR3 foi adicionado e incubado em temperatura ambiente por 2 horas. O anticorpo ligado especificamente a FGFR3 foi detectado usando um anti-Fab humano conjugado com HRP e substrato cromogênico TMB peroxidase (KPL, Gaithersburg, MD).
[525] Para testar o efeito de anticorpos para FGFR3 sobre a interação FGF/FGFR3, as proteínas quiméricas FGFR3-Fc foram capturadas em placas Maxisorp revestidas com anticorpo anti-imunoglobulina FCY humana fragmento-específico (Jackson Immunoresearch, West Grove, PA). Após a lavagem, uma quantidade crescente de anticorpos FGFR3 foi adicionada à placa e incubada por 30 minutos. Então, FGF1 ou FGF9 e heparina foram adicionadas para a incubação à temperatura ambiente por 2 horas. As placas foram lavadas e incubadas por uma hora com o anticorpo policlonal específico para FGF1 biotinilado (BAF232) ou anticorpo para FGF9 biotinilado (BAF273 R&D Systems), seguido pela detecção com estreptavidina-HRP e TMB.GERAÇÃO DE CÉLULAS BA/F3-FGFR3 ESTÁVEIS
[526] O cDNA codificando o FGFR3 IIIb e IIIc humano de comprimento total foi clonado no vetor pQCXIP (Clontech Laboratories, Mountain View, CA) para gerar o pQCXIP-FGFR3-IIIb e -IIIc. Mutações específicas, ou seja, R248C, S249C, G372C, Y375C e K652E, foram introduzidas no cDNA utilizando o QuickChange (Stratagene, La Jolla, CA). Para gerar células Ba/F3 estáveis expressando o FGFR3 tipo selvagem ou mutante, várias construções pQCXIP - FGFR3 foram co-transfectadas em células GP2-293 com o plasmídeo VSV -G (Clontech Laboratories). Após a seleção com 2 μh/mL de puromicina por duas semanas, as células que expressam FGFR3 tipo selvagem ou mutante foram coradas com anticorpo monoclonal (mAb) anti-FGFR3 humano conjugado com Ficoeritrina (FAB766P, R&D Systems), e selecionados por meio da técnica de separação de células ativadas por fluorescência (FACS) para ensaios funcionais. Para o ensaio de proliferação celular em placas de micro-titulação de 96 poços, a densidade de células utilizada foi a seguinte: Para células que expressam FGFR3-IIIb tipo selvagem e FGFR3-K652E: 5.000 células/poço, para o resto: 10.000 células/poço. As células foram semeadas em meio RPMI-1640 suplementado com 10% de soro bovino fetal, 10 ng/ml de FGF1 mais 10 μg/ml de heparina (Sigma -Aldrich, St. Louis, MO). O R3Mab foi adicionado na concentração indicada e anticorpos FGFR3 de hibridoma de camundongo foram adicionados de 2000 a 0,49 ng/ml (em diluições seriadas de quatro vezes) no experimento com FGFR3-IIIb e 5000 a 1,2 ng/ml (em diluições seriadas de quatro vezes) no experimento com FGFR3-IIIc. Após incubação por 72 horas, a viabilidade celular foi avaliada utilizando o CellTiter -Glo (Promega, Madison, WI).ENSAIO DE PROLIFERAÇÃO CELULAR
[527] Para ensaios de proliferação para células RT112, RT4 e TCC- 97-7, foram semeadas 3000 células/poço em placas de microtitulação de 96 poços e as células foram deixadas em repouso durante a noite para se aderirem. O meio foi então substituído por meio com baixa concentração de soro (0,5% de FBS), com controle ou R3Mab nas concentrações indicadas. Após quatro dias de incubação, 1 μCi de [metil-H3]-timidina (PerkinElmer, Waltham, MA) foi adicionado a cada poço e incubado por mais 16 horas. As células foram transferidas para UniFilters usando o aparato a coleta Packard Filtermate Harvester, e a [H3] timidina incorporada ao DNA genômico das células em crescimento foi mensurada utilizando o TopCount (PerkinElmer). Em alguns casos, a viabilidade celular foi avaliada com o CellTiter-Glo (Promega), após incubação com os anticorpos por 4 dias. Os valores são apresentados como média +/- EP de quadruplicatas.ENSAIO DE CRESCIMENTO CLONAL
[528] O efeito do R3Mab sobre a clonogenicidade celular foi avaliado de acordo com um protocolo descrito previamente (50). Resumidamente, 400 células UMUC-14 foram semeadas em placas de 6 poços em meio DMEM suplementado com 10% de soro fetal bovino para permitir a aderência durante a noite. Em seguida anticorpos R3Mab ou controle diluídos em meio com 0,1% de BSA foram adicionados a uma concentração final de 10 μg/ml. Volumes iguais de médio sozinho com 0,1% de BSA (Mock) foram usados como outro controle. As células foram incubadas por cerca de 12 dias até que as células nos grupos controle formaram colônias suficientemente grandes. As colônias foram coradas com cristal violeta a 0,5%, e o número e tamanho das colônias foram quantificados usando o GelCount (Oxford, UK). O número de colônias maiores que 120 μm de diâmetro foram apresentados como média +/- EPM (n = 12).ANÁLISES DE IMUNOPRECIPITAÇÃO E IMMUNOBLOTTING
[529] Para estudar o efeito de anticorpos sobre a sinalização de FGFR3, as células foram privadas de nutriente em meio sem soro durante a noite antes do início do tratamento. As células foram incubadas com anticorpos ou diluídas em BSA a 0,1% (p/v), meio RPMI-1640, ou apenas meio com 0,1% de BSA (Mock). Após 3 horas a 37 °C, FGF1 (concentração final de 15 ng/ml) e heparina (concentração final de 50-10 μg/ml) foram adicionados à metade das amostras. Como controles, um volume similar de heparina isoladamente foi adicionado à outra metade das amostras. A incubação foi mantida por 10 min. Os sobrenadantes foram removidos por aspiração, e as células foram lavadas com PBS gelado, então lisadas em tampão RIPA (Upstate, Charlottesville, VA) suplementado com 1 mM de ortovanadato de sódio e coquetel inibidor de protease completo (Roche Applied Science, Indianapolis, IN). Os lisados foram depurados de materiais insolúveis por centrifugação.
[530] O FGFR3 foi imunoprecipitado usando um anticorpo policlonal de coelho (sc-123, Santa Cruz Biotechnology, Santa Cruz, CA) e analisado por eletroforese em gel de dodecil-poliacrilamida (SDS- PAGE) e Western blot. O FGFR3 fosforilado foi avaliado com um anticorpo monoclonal contra fosfotirosina (4G10, Upstate). O FGFR3 total foi sondado com um anticorpo monoclonal contra FGFR3 (sc- 13121, Santa Cruz Biotechnology). A fosforilação e ativação da via de sinalização do FGFR3 foram sondadas usando os anticorpos: anti-FGFRY653/654, anti-FRS2αY196, anti-fosfo-p44/42MAPKT202/Y204, anti-p44/42 MAPK total e anti-AKTS473 foram obtidos a partir da Cell Signaling Technology (Danvers, MA); e anti-FRS2α total (sc-8318) foi comprado da Santa Cruz Biotechnology (Santa Cruz, CA). Os blots foram visualizados por meio de um substrato quimioluminescente (ECL Plus, Amersham Pharmacia Biotech, Piscataway, NJ).MAPEAMENTO DE EPÍTOPOS DE ANTICORPOS
[531] Para determinar o epítopo de R3Mab, 13 de peptídeos sobrepostos, cada um com 15 aminoácidos de comprimento, foram sintetizados para cobrir o domínio extracelular do FGFR3 humano a partir do resíduo 138 até o 310. Os peptídeos foram biotinilados na extremidade C-terminal, e capturados em placas com estreptavidina (Pierce, Rockford, IL) durante a noite. Após bloqueio com PBS/BSA 3%, as placas foram incubadas com R3Mab e detectado através de um anti-IgG humano conjugado com HRP (Immunoresearch Jackson) e o substrato cromogênico TMB peroxidase (KPL, Gaithersburg, MD).
[532] Anticorpo de camundongo anti-FGFR3 de hibridoma humano 1G6, 6G1 e 15B2 foram testados no teste de ELISA para identificar os seus epítopos de ligação. 1G6, 6G1 e 15B2 se ligam ao IgD2-IgD3 FGFR3 humano (ambas isoformas IIIb e IIIc), enquanto que 5B8 se liga apenas ao IgD2- IgD3 de FGFR3-IIIb humano. Em um ensaio de competição, 1G6, 6G1 e 15B2 competiam entre si para se ligarem ao FGFR3 humano, sugerindo que 1G6, 6G1 e 15B2 possuem epítopos sobrepostos. Nenhum dos anticorpos de hibridoma competiu com o anticorpo fago 184,6, sugerindo que os anticorpos de hibridoma possuem epítopos distintos do 184,6.PREPARAÇÃO E CLONAGEM MOLECULAR DE ANTICORPOS CAMUNDONGO ANTI- FGFR3 1G6, 6G1 E 15B2
[533] Camundongos BALB/c foram imunizados 12 vezes com 2,0 μg de FGFR3-IIIb rhFGFR3 (IIIB)/Fc quimérico, da R&D Systems, catalogo 1264- FR, lote # CYH025011, ou com 2,0 μg de FGFR3-IIIc rhFGFR3 (IIIc)/Fc quimérico, da R&D Systems, catalogo 766-FR, lote #CWZ055041, ressuspensos em monofosforil lipídio A/adjuvante dicorinomicolato de trealose (Corixa, Hamilton, MT) em cada pata traseira duas vezes por semana. Três dias após a imunização intensificadora final, os linfonodos poplíteos foram fundidos com células de mieloma de camundongo da linhagem P3X63Ag.U.1 através de eletrofusão (Hybrimune, Cyto Pulse Sciences, Glen Burnie, Maryland). As células de hibridoma fusionadas foram selecionadas a partir do linfonodo poplíteo não fundido ou de células do mieloma utilizando a seleção por hipoxantina-aminopterina-timidina (HAT) em meio D do kit de seleção “ClonaCell hibridoma selection kit” (StemCell Technologies, Inc., Vancouver, BC, Canadá). Os sobrenadantes da cultura foram examinados inicialmente por suas capacidades de se ligarem a FGFR3-IIIb e FGFR3-IIIc por ELISA, e os hibridomas de interesse foram posteriormente selecionados por suas capacidades de coloração por FACS em células Ba/F transfectadas com FGFR3- IIIb e células Ba/F controle, bem como a atividade bloqueadora dos anticorpos. Os hibridomas selecionados foram então clonados por diluição limitante.
[534] O RNA total foi extraído das células de hibridoma produzindo o anticorpo monoclonal de camundongo anti-FGFRIII humano 1G6 e 15B2, utilizando o kit de extração “RNeasy Mini Kit” (Qiagen, Alemanha). Os domínios de cadeia leve (VK) e pesada (VH) foram amplificados por RT-PCR com os seguintes primers degenerados:1G6:Cadeia Leve (LC) direto (forward): 5’-GTCAGATATCGTKCTSACMCARTCTCCWGC-3’ (SEQ ID NO:195)Cadeia pesada (HC) direto (forward):5’-GATCGACGTACGCTGAGATCCARYTGCARCARTCTGG-3’(SEQ ID NO:196)6G1:Cadeia Leve (LC) direto (forward): 5’-GTCAGATATCGTGCTGACMCARTCTCC-3’ (SEQ ID NO:197)Cadeia pesada (HC) direto (forward): 5’-GATCGACGTACGCTGAGATCCARYTGCARCARTCTGG-3’ (SEQ ID NO:198)15B2:Cadeia Leve (LC) direto (forward): 5’-GTACGATATCCAGATGACMCARTCTCC-3’ (SEQ ID NO:199)Cadeia pesada (HC) direto (forward): 5’-GATCGACGTACGCTGAGATCCARYTGCARCARTCTGG-3’ (SEQ ID NO:200)Cadeia Leve e Cadeia pesada reverso (reverse) primer para os três clones são os seguintes:Cadeia Leve reverso (reverse): 5’-TTTDAKYTCCAGCTTGGTACC-3’ (SEQ ID NO:201)Cadeia pesada reverso (reverse): 5’-ACAGTGGGCCCTTGGTGGAGGCTGMRGAGACDGTGASHRDRGT-3’ (SEQ ID NO:202).
[535] Os primers diretos (forward) eram específicos para a sequência de aminoácido N-terminal da região VL e VH. Os primers reversos LC e HC foram desenhados para anelarem a uma região no domínio constante leve (CL) e constante pesado 1 (CH1), respectivamente, que são altamenteconservados entre as espécies.
[536] O VL amplificado foi clonado em um vetor de expressão de células de mamíferos pRK (Shields et al., (2000) J. Biol. Chem. 276:659) contendo o domínio constante kappa humano. O VH amplificado foi inserido em um vetor de expressão de células de mamíferos pRK codificando o domínio constante do IgG1 humano de comprimento total. A sequência das cadeias pesadas e leves foi determinada utilizando métodos convencionais.CRISTALIZAÇÃO, DETERMINAÇÃO DA ESTRUTURA E REFINAMENTO
[537] O ECD FGFR3-IIIb humano (resíduos 143-374) foi clonado no vetor pAcGP67A (BD Bioscience, San Jose, CA), produzido em células T.ni Pro e purificado utilizando uma coluna Ni-NTA seguido por cromatografia de exclusão de tamanho. O Fab R3Mab foi expresso em E. coli e purificado sequencialmente em uma coluna de afinidade de proteína G, uma coluna de SP sepharose e uma coluna Superdex 75. O complexo Fab - FGFR3 foi gerado pela incubação do Fab com um excesso de ECD FGFR3, e o complexo foi então deglicosilado e purificado através de uma coluna de exclusão Superdex-200 em 20 mM de TrisCl pH 7,5 e 200 mM de tampão NaCl. As frações contendo complexo foram reunidas e concentradas a 20 mg/ml e utilizadas nos ensaios de cristalização. Os cristais utilizados na determinação da estrutura foram mantidos a 4 °C nas seguintes condições: 0,1 M de cacodilato de sódio pH 6,5, MPD 40% e PEG8000 5% utilizando o método de difusão a vapor. Os dados foram processados usando o HKL2000 e Scalepack (56). A estrutura foi resolvida com a substituição molecular utilizando o programa Phaser (57) e as coordenadas de 1RY3 (FGFR3) e 1N8Z (fragmento Fab). O modelo foi concluído usando o programa Coot (58) e a estrutura aperfeiçoada a um R/Rfree de 20,4%/24,3% com programa Refmac (59). Fatores de coordenadas e estrutura foram depositados no Banco de Dados de Proteína com o código de acesso 3GRW e também divulgadas no USSN 61/163.222, depositado em 25 de março de 2009, cujo conteúdo é incorporado apresente pela referência.ENSAIO DE ADCC
[538] PBMCs humanas foram isoladas por centrifugação em gradiente de Ficoll de sangue heparinizado, e a ADCC foi mensurada usando as linhagens de células de mieloma múltiplo OPM2 ou KMS11 ou linhagens de células de câncer de bexiga RT112 ou UMUC-14 como alvos e as PBMCs como células efetoras em uma relação célula alvo:célula efetora de 1:100. As células- alvo (10.000 células/poço) foram tratadas com R3Mab ou com IgG1 humano como controle por 4 horas a 37 °C. A citotoxicidade foi determinada mensurando a liberação de LDH usando o ensaio “CytoTox-ONE Homogeneous Membrane Integrity Assay” seguindo as instruções do fabricante (Promega, Madison, WI). Os resultados são expressos em porcentagem de citólise específica usando a fórmula: Citotoxicidade (%) = [(lise experimental - lise experimental espontânea) / (lise máxima das células alvo - lise espontânea das células alvo)] x 100, onde a lise espontânea é a citólise inespecífica na ausência de anticorpos, e lise de células alvo máxima é induzida por 1% de Triton X-100.RESULTADOSSlLENCIAMENTO GÊNICO (KNOCKDOWN) POR SHRNA INDUZÍVEL DE FGFR3 ATENUA O CRESCIMENTO DO CÂNCER DE BEXIGA EM EXPERIMENTO IN VIVO
[539] Como um prelúdio para avaliar a importância do FGFR3 no crescimento do tumor de bexiga in vivo, os inventores examinaram o efeito de um silenciamento gênico (knockdown) do FGFR3 em um ensaio in vitro. Diversos RNAs interferentes pequenos (siRNA) de FGFR3 regularam negativamente as linhagens de células de câncer de bexiga que expressavam FGFR3 tanto do tipo selvagem (WT) (RT112, RT4, SW780) quanto mutante (UMUC-14, mutação S249C). O silenciamento gênico de FGFR3 em todas as quatro linhagens de células suprimiu evidentemente a proliferação em cultura (Figura 15). Em seguida, foram geradas as linhagens de RT112 expressando shRNA de FGFR3 induzível por doxiciclina. A indução de três shRNAs de FGFR3 independentes por doxiciclina diminuiu a expressão de FGFR3, ao passo que a indução de um shRNA controle direcionado para EGFP não teve efeito (Figura 7A). Na ausência de FGF exógeno, o tratamento com doxiciclina reduziu a incorporação de [H3]- timidina pelas células que expressam diferentes shRNAs de FGFR3, mas não as que expressavam shRNA controle (Figura 7B), confirmando que o silenciamento gênico de FGFR3 inibe a proliferação. Análise mais aprofundada das células em crescimento exponencial RT112 revelou que o silenciamento gênico de FGFR3 durante um tratamento de 72 hr com doxiciclina reduziu nitidamente e especificamente a porcentagem de células nas fases S e G2 do ciclo celular, com um aumento concomitante de células na fase G1 (Figura 7C). Efeitos similares foram observados com outros dois shRNAs FGFR3 (Figura 16a). Não houve um número significativo de células com um conteúdo de DNA sub- diploide, sugerindo que não há alteração nos níveis de apoptose. Assim, o efeito inibitório do silenciamento gênico de FGFR3 sobre a proliferação de células RT112 é principalmente devido à atenuação da progressão do ciclo celular.
[540] Os inventores avaliaram o efeito do silenciamento gênico FGFR3 no crescimento de tumores xenoenxertados pré-estabelecidos de células RT112 em camundongos. O silenciamento gênico de FGFR3 suprimiu substancialmente e especificamente o crescimento do tumor (Figura 7D, painéis superiores e Figura 16B). A análise das amostras de tumores do dia 45 confirmou a eficácia do silenciamento gênico do FGFR3 após a indução do shRNA para FGFR3 com doxiciclina em comparação com o shRNA controle (Figura 7D, painéis inferiores). Estes resultados demonstram que FGFR3 é extremamente importante tanto in vitro como in vivo para o crescimento de células câncer de bexiga RT112.GERAÇÃO DE UM ANTICORPO MONOCLONAL ANTI-FGFR3 DE BLOQUEIO
[541] Para analisar a importância do FGFR3 no crescimento tumoral e explorar o potencial deste receptor como alvo terapêutico desenvolveu um anticorpo monoclonal anti-FGFR3 antagônico (apelidado de R3Mab) usando uma abordagem de exibição por fago. Selecionamos este anticorpo específico com base em sua capacidade de bloquear tanto a ligação como a dimerização pelo FGFR3, e sua capacidade única para inibir não apenas FGFR3 WT como também os as formas mutantes mais prevalentes deste receptor associadas ao câncer (vide abaixo). O R3Mab é direcionado aos domínios extracelulares IgD2 e IgD3 do FGFR3, que são necessários e suficientes para ligação ao FGF (4). O R3Mab se liga a ambas isoformas IIIb e IIIc do FGFR3 humano, mas não exibem nenhuma ligação detectável para FGFR1, FGFR2 ou FGFR4 (Figura 8A). A análise do R3Mab por Biacore indicou que este anticorpo possui afinidade semelhante ao FGFR3-IIIc murino, de macacos cinomolgos e de humanos (dados não mostrados). A afinidade do R3Mab para o FGFR3 humano é exibida na Tabela 2.
[542] A seguir, foi testada a capacidade do R3Mab bloquear aligação do FGFR3 ao FGF1 e FGF9. O R3Mab inibiu fortemente a ligação doFGF1 ao FGFR3-IIIb e -IIIc, com um valor de Concentração Inibidora a 50% (IC50) de 0,3 nM e 1,7 nM, respectivamente (Figura 8B, C). Da mesma forma, R3Mab bloqueia eficientemente a ligação do FGF9 ao FGFR3-IIIb e -IIIc com um valor IC50 de 1,1 nM e 1,3 nM, respectivamente (Figura 8D, E).R3MAB INIBE FGFR3 WT E SEUS VARIANTES MUTANTES MAIS PREVALENTEMENTE ASSOCIADOS AO CÂNCER
[543] Para examinar se R3Mab inibe a proliferação celular direcionada pelo FGFR3 WT ou mutante aproveitou a observação de que a expressão ectópica de FGFR3 em célula pró-B murina Ba/F3 confere proliferação e sobrevida independente de interleucina (IL)-3, e dependente de FGF1 (29). Na ausência de FGF1 e IL-3, as células Ba/F3 expressando estavelmente FGFR3 WT não eram viáveis, enquanto o FGF1 melhorava bastante a proliferação destas (Figura 9A). O R3Mab bloqueou especificamente a proliferação de células Ba/F3-FGFR3 estimuladas por FGF1 de uma forma dose-dependente (Figura 9A). A seguir, avaliou-se o impacto do R3Mab na sinalização de FGFR3 nessas células. O FGF1 induziu a fosforilação e ativação de FGFR3 e ativação concomitante da p44/42 MAPK, enquanto R3Mab suprimiu efetivamente a ativação de ambas as moléculas (Figura 9B).
[544] No câncer de bexiga, mutações somáticas ativadoras no FGFR3 agrupam-se dentro da região ligante entre IgD3 e IgD2, o domínio justamembrana extracelular ou o domínio quinase (Figura 9C). As substituições do tipo missense extracelulares dão origem na maioria das vezes a uma cisteína não pareada, levando a dimerização de FGFR3 de maneira independente de ligante. Essas mutações causam níveis muito diferentes de ativação constitutiva do FGFR3, possivelmente devido a um impacto diferencial sobre a orientação do domínio quinase citoplasmático (30, 31). As mutações mais frequentes são S249C, Y375C, R248C, G372C e K652E, que juntas respondem por 98% de todas as mutações de FGFR3 no câncer de bexiga (32). Desse, modo os inventores avaliaram que um agente terapêutico ideal deveria bloquear não só a proteína FGFR3 do tipo selvagem, que é abundante em certos tipos de câncer, como também as formas de FGFR3 mutantes mais prevalentemente associadas aos tumores. Para avaliar esse efeito do R3Mab, foram geradas linhagens de células Ba/F3 expressando estavelmente cada uma das cinco variantes de FGFR3 mutantes mais comuns. Todos os mutantes foram expressos em níveis semelhantes na superfície da célula, e os mutantes cisteína dimerizavam espontaneamente sem ligantes (dados não mostrados). As diferentes linhagens de células mutantes expressando cisteína exibiram uma resposta de crescimento variável para FGF1, consistente com os achados anteriores (30, 31). Como relatado anteriormente (33), as células que expressavam FGFR3R248C exibiam proliferação constitutiva independente de ligante e eram não responsivas ao FGF1 (Figura 9D). Da mesma forma, a mutação mais frequente, FGFR3S249C, conferiu uma proliferação independente de ligante (Figura 9E). De forma surpreendente, o R3Mab suprimiu a proliferação constitutiva induzida por ambos mutantes (Figura 9 D, E). As Células que expressam mutações no domínio justamembrana FGFR3G372C (Figura 9F) ou FGFR3Y375C (Figura 9G) necessitam de FGF1 para a proliferação, e seu crescimento foi completamente bloqueado pelo R3Mab. As células expressando FGFR3K652E exibiram fraca proliferação independente de ligante e um significante crescimento em resposta a FGF1 (33). O R3Mab não afetou a fraca atividade basal de FGFR3K652E (dados não mostrados), mas quase aboliu a proliferação induzida por ligante mediada por esse mutante (Figura 9H). Assim, R3Mab tem uma capacidade única para inibir tanto o FGFR3 WT quanto as formas de FGFR3 mutantes mais prevalentes associadas ao câncer. Além disso, R3Mab não apresenta atividade agonista detectável.
[545] Como um esforço separado, os inventores geraram e caracterizaram vários anticorpos de hibridoma camundongo anti-FGFR3 humano. Nenhum dos anticorpos de hibridoma pode inibir todas as formas mutantes de FGFR3 associadas aos cânceres testados (Figura 17), e nem compartilharam a sobreposição de epítopos com o R3Mab.
[546] Além disso, todos os anticorpos de hibridoma exibiram atividade agonista, estimulando fortemente a proliferação de FGFR3 mutantes associados ao câncer R248C e S249C, e mostraram alguma estimulação sobre a proliferação em mutantes Y375C e G370C. Os anticorpos de hibridoma apresentaram os seguintes níveis diferenciais de antagonismo e agonismo, dependendo do mutante FGFR3 testado:
[547] Assim, os anticorpos de hibridoma exibiram efeitosdiferenciais imprevisíveis sobre a proliferação celular de células Ba/F3 induzidas por vários mutantes de FGFR3.CARACTERIZAÇÃO DE ANTICORPOS DE HIBRIDOMA CAMUNDONGO ANTI-FGFR3 HUMANO
[548] Os anticorpos de hibridoma camundongo anti-FGFR3 humano foram adicionalmente caracterizados da seguinte forma:(1) Em um ensaio para testar a capacidade dos anticorpos anti- FGFR3 de hibridoma murino inibirem a ligação do FGF1 às isoformas de FGFR3- IIIb e -IIIc humano, os anticorpos 1G6, 6G1 e 15B2 foram capazes de bloquear a ligação do FGF1 ao FGFR3 humano isoformas -IIIb e -IIIc de uma forma dose- dependente. Quando testados em uma faixa de concentração de anticorpos de cerca de 2.000 a 0,49 ng/ml, os anticorpos 1G6, 6G1 e 15B2 bloquearam a ligação do FGF1 ao FGFR3-IIIb com valores IC50 de 0,69, 0,87 e 0,72 nM. Quando testados em uma faixa de concentração de anticorpos de cerca de 5.000 a 1,2 ng/ml, os anticorpos 1G6, 6G1 e 15B2 bloquearam a ligação do FGF1 ao FGFR3-IIIc com valores IC50 de 0,57, 3,4 e 0,7 nM;(2) Em um ensaio para testar a capacidade dos anticorpos anti- FGFR3 de hibridoma murino inibirem a ligação do FGF9 ao FGFR3 humano isoformas -IIIb e -IIIc, os anticorpos 1G6, 6G1 e 15B2 bloquearam eficientemente a ligação do FGF1 ao FGFR3 humano isoformas -IIIb e -IIIc de uma maneira dose-dependente. Quando testados em uma faixa de concentração de anticorpos de cerca de 2.000 a 0,49 ng, os anticorpos 1G6, 6G1 e 15B2 bloquearam a ligação do FGF9 ao FGFR3-IIIc com valores IC50 de 0,13, 0,16 e 0,07 nM, respectivamente. Quando testados em uma faixa de concentração de anticorpos de cerca de 5.000 a 1,2 ng/ml, os anticorpos 1G6, 6G1 e 15B2 bloquearam a ligação do FGF9 ao FGFR3-IIIc com valores IC50 de 0,13, 0,11 e 0,07 nM, respectivamente;(3) A afinidade de ligação dos anticorpos de hibridoma murinos anti-FGFR3 de comprimento total 1G6, 6G1 e 15B2 foi determinada usando a análise Biacore. Os resultados desta análise são apresentados na Tabela 3;(4) Em um ensaio para testar a capacidade dos anticorpos anti-FGFR3 de hibridoma murino inibirem a proliferação de células Ba/F3 induzida pelo FGFR3-IIIb e -IIIc humano, os anticorpos 1G6, 6G1 e 15B2 foram capazes de bloquear a proliferação de células Ba/F3 induzida pelo FGFR3 humano isoformas -IIIb e -IIIc de maneira dose-dependente. Quando testado em uma faixa de concentração de anticorpos de cerca de 0,01 a 100 μg/ml, os anticorpos 1G6, 6G1 e 15B2 bloquearam a proliferação celular de células Ba/F3 induzida por FGFR3-IIIb com valores IC50 de 3-5 nM, 3 nM, e 6-8 nM, respectivamente, e bloqueou a proliferação celular de Ba/F3 induzida por FGFR3 - IIIc, com valores IC50 de 10-35 nM, 24 nM e 60 nM, respectivamente;[5] Em um ensaio para testar a capacidade dos anticorpos de hibridoma murino anti-FGFR3 inibirem a sinalização induzida por FGF1 em células BA/F3 que expressam FGFR3-IIIb humano, os anticorpos 1G6, 6G1 e 15B2 foram capazes de bloquear a sinalização induzida por FGF1 em células Ba/F3 expressando FGFR3-IIIb humano de forma dose-dependente, quando testados em uma faixa de concentração de anticorpos de cerca de 0,25 - 6,75 μg/ml. 25 ng/ml de FGF1 foi utilizado neste experimento. Na ausência de FGF1, o tratamento de anticorpos não teve efeito sobre a ativação de FGFR3;[6] Em um ensaio para testar a capacidade dos anticorpos de hibridoma murino anti-FGFR3 inibirem a sinalização induzida por FGF1 em células BA/F3 que expressam FGFR3-IIIc humano, os anticorpos 1G6, 6G1 e 15B2 foram capazes de bloquear a sinalização induzida por FGF1 em células Ba/F3 expressando FGFR3-IIIc humano de forma dose-dependente, quando testados em uma faixa de concentração de anticorpos de cerca de 0,25 - 6,75 μg/ml. 25 ng/ml de FGF1 foi utilizado neste experimento. Na ausência de FGF1, o tratamento de anticorpos não teve efeito sobre a ativação de FGFR3.[7] E ESTRUTURAL PARA A INTERAÇÃO DE R3MAB COM FGFR3
[549] Para compreender sobre o modo que o R3Mab interage com o FGFR3, os inventores sintetizaram um painel de 13 peptídeos sobrepostos que abrangem as regiões IgD2 e D3 do FGFR3-IIIb e testaram sua ligação ao R3Mab. Os peptídeos 3 (resíduos 164-178) e 11 (resíduos 269-283) exibiram ligação específica para o R3Mab, sendo que o peptídeo 3 exibiu uma interação mais forte (Figura 10A), indicando que as regiões correspondentes no FGFR3 são críticas para o reconhecimento por parte do R3Mab. Estudos anteriores de cristalografia do FGFR1 no complexo com FGF2 identificaram resíduos receptores fundamentais envolvidos na ligação direta ao FGF e heparina, bem como na dimerização do receptor (34). O alinhamento dos peptídeos 3 e 11 FGFR3 com os sítios funcionalmente importantes no FGFR1 revelou que estes peptídeos abrangem resíduos do FGFR1 correspondentes essenciais para a ligação direta de FGF2, dimerização do receptor, bem como a interação com a heparina (Figura 10B). Estes dados indicam que o epítopo de R3Mab sobre FGFR3 se sobrepõem com os resíduos do receptor envolvidos na associação ligante e interação receptor-receptor.
[550] Em seguida foi cristalizado o complexo entre o fragmento Fab do R3Mab e a região IgD2-D3 extracelular do FGFR3-IIIb humano, e determinou-se a estrutura de raios-X em resolução de 2,1A (Figura 10 C, D; Tabela 4). Neste complexo, aproximadamente 1400 A2 a 1500 A2 de áreas acessíveis da superfície acessíveis ao solvente estão encobertas no FGFR3 e o Fab, respectivamente. Cerca de 80% da interface encoberta envolve IgD2, enquanto o restante envolve as regiões ligante e IgD3. No lado Fab do complexo, cerca de 40% da interface encoberta envolvem a região determinante de complementaridade (CDR)-H3, 20% CDR-H2, 20% CDR-L2, e contribuições menores são de outras CDRs e resíduos da região estrutural. Notavelmente, os aminoácidos (AAs) do CDR-H3 formam duas fitas-β, que se estendem as folhas- β da IgD2 (Figura 10D). O Fab interage com os AAs que constituem o sítio de ligação ao FGF do FGFR3, bem como resíduos que formam a interface de dimerização do receptor, como já identificado em vários complexos diméricos FGF:FGFR (por exemplo, PDB código 1CVS, (34); e Figura 10C, áreas em cinza/hachuradas e cinza escuro). As interfaces de interação identificadas por cristalografia foram totalmente consistentes com os dados baseados nos peptídeos (Figura 18 A, B). Juntos, estes resultados revelam como R3Mab inibe a ligação do ligante, e adicionalmente sugerem que a ligação do R3Mab para o FGFR3 pode impedir dimerização do receptor. Os aminoácidos FGFR3 que contatam R3Mab são exibidos na Tabela 5. As coordenadas cristalográficas para esta estrutura estão depositadas no Baco de Dados de Proteína (Protein Data Bank) com código de acesso 3GRW.aRsym = ∑|I-<I>| / ∑ I. <I> é a intensidade média das observações relacionadas com a simetria de uma reflexão única. b Números em parênteses referem-se à camada de mais alta resolução. cR = ∑|Fo-Fc| / ∑Fo. Rfree é calculado como R, mas para 5% das reflexões excluídas de todos os refinamentos.
[551] Os inventores compararam a estrutura R3Mab-FGFR3 com uma estrutura previamente publicada do FGFR3-IIIc em complexo com FGF1 (4, 35) (Figura 10E, 10F). A superposição dos complexos anticorpo-receptor e ligante-receptor revelou que não existem grandes diferenças conformacionais dentro dos domínios do receptor individual, exceto na região que distingue FGFR3-IIIc de FGFR3-IIIb, no entanto, a orientação do IgD3 em relação ao IgD2 foi drasticamente diferente (Figura 10E, branco e cinza; Figura 10F, branco e cinza - em textura). Uma vez que as posições relativas de IgD2 e IgD3 são críticas para a ligação do ligante, a conformação alternativa adotada pela IgD3 sobre a ligação de R3Mab pode fornecer um mecanismo adicional para evitar a interação do ligante com FGFR3.R3MAB INIBE FGFR3 ENDÓGENO WT E MUTANTE EM CÉLULAS DE CÂNCER DE BEXIGA
[552] Para avaliar se R3Mab poderia suprimir a função do FGFR3 em células de câncer de bexiga, os inventores examinaram primeiramente as linhagens de células RT112 e RT4, que expressam FGFR3 WT. O R3Mab inibiu fortemente a incorporação de [H3]-timidina pelas células RT112 (Figura 11a) e exerceu uma supressão significativa, embora mais moderada, sobre a proliferação das células RT4 (Figura 19a). Para investigar o efeito do R3Mab sobre a ativação de FGFR3, os inventores examinaram a fosforilação de FGFR3 em células RT112. Consistentes com os resultados obtidos com as células Ba/F3-FGFR3 (Figura 9B), o R3Mab atenuou nitidamente a fosforilação de FGFR3 induzida por FGF1 (Figura 11B). A seguir, os inventores examinaram a fosforilação de FRS2α, AKT e MAPK p44/42, três mediadores a jusante da sinalização de FGFR3. O FGF1 ativou fortemente essas moléculas nas células RT112, enquanto o R3Mab diminuiu significativamente esta ativação (Figura 11B). Da mesma forma, R3Mab suprimiu a fosforilação de FGFR3 e MAPK induzida por FGF1 em células RT4 (Figura 19B).
[553] Em seguida, os inventores investigaram se o R3Mab poderia inibir a ativação do FGFR3 mutante endógeno em células humanas de câncer de bexiga. A S249C é a mutação de FGFR3 mais frequentes no câncer de bexiga (Figura 9C). Duas linhagens de células disponíveis, UMUC -14 e TCC- 97-7, carregam um alelo FGFR3S249C mutado (Ref. 36 e dados não mostrados). Embora o R3Mab não afetou o crescimento exponencial das células UMUC -14 em cultura (dados não mostrados), ele reduziu significativamente o crescimento clonal dessas células (Figura 11C). Especificamente, o R3Mab diminuiu o número de colônias maiores que 120 μm de diâmetro em aproximadamente 77% em comparação com o anticorpo controle (Figura 11D). Além disso, o R3Mab inibiu a incorporação de [H3]-timidina pelas células TCC- 97-7 em cultura (Figura 19C).
[554] A mutação S249C é descrita por resultar na ativação do FGFR3 independente de ligante (26, 30). Na verdade, o FGFR3S249C foiconstitutivamente fosforilado independentemente do tratamento com FGF1 nas células UMUC-14 e TCC-97-7, enquanto o R3Mab reduziu a fosforilação constitutiva do FGFR3S249C em comparação com o anticorpo controle em ambas as linhagens de células (Figuras 11E, 19D).R3MAB INIBE A FORMAÇÃO DE DÍMERO PELO FGFR3S249C
[555] A capacidade do R3Mab de inibir a sinalização constitutiva do FGFR3S249C e a proliferação de células de câncer de bexiga foi surpreendente, considerando que este mutante pode sofrer ligações de dissulfeto, e dimerização independente de ligante (26, 30). Para explorar como R3Mab inibe FGFR3S249C, os inventores examinaram o efeito do R3Mab sobre o estado oligomérico deste mutante em células UMUC -14. Sob condições redutoras, o FGFR3S249C migrou como uma banda única de ~ 97 kDa, compatível com o tamanho monomérico (Figura 12A). Sob condições não-redutoras, em células tratadas com o anticorpo controle uma grande fração de FGFR3S249C apareceu como uma banda de ~ 200 kDa, independentemente da adição de FGF1, indicando um estado dimérico constitutivo (Figura 12A). O tratamento com R3Mab diminuiu substancialmente a quantidade de dímeros, com um aumento concomitante de monômeros (Figura 12A). Consistentemente, o R3Mab diminuiu o nível de dímeros FGFR3S249C em células TCC- 97-7, independentemente do tratamento com FGF1 (Figura 19E).
[556] Como o R3Mab diminui os níveis do dímero FGFR3S249C nas células de câncer de bexiga? Uma possível explicação é que ele pode perturbar o dímero FGFR3S249C por meio da internalização de FGFR3 induzida por anticorpo e tráfico através de endossomos ou lisossomos. Essa possibilidade foi avaliada farmacologicamente pela intervenção da endocitose. No entanto o R3Mab diminuiu a quantidade de dímeros em células UMUC-14 pré- tratadas com diversos inibidores da endocitose, apesar do bloqueio substancial da internalização de FGFR3S249C (Figura 20 A, B). Assim, a interrupção do dímero pelo R3Mab é independente da endocitose. Outra possível explicação é que o FGFR3S249C celular pode existir em um equilíbrio dinâmico dímero-monômero e, dessa forma, a ligação do R3Mab ao FGFR3S249C monomérico poderia impedir a formação de dímeros e, assim, deslocar o equilíbrio para o estado monomérico. Ao examinar essa possibilidade, utilizou-se o agente permeante não celular ácido 5,5-ditiobis-2-nitrobenzóico (DTNB), que reage seletivamente e bloqueia grupos de sulfidrila livres de cisteínas não pareadas (37). O tratamento de células UMUC-14 com DTNB levou ao acúmulo de monômeros FGFR3S249C à custa de dímeros (Figura 12B), indicando que o FGFR3S249C existe em um equilíbrio dinâmico entre monômeros e dímeros.
[557] Para avaliar se R3Mab afeta esse equilíbrio, geramos uma proteína recombinante solúvel compreendendo os domínios IgD2-D3 do FGFR3S249C e os dímeros foram isolados por cromatografia por exclusão de tamanho. Os dímeros foram incubados com tampão ou anticorpos na presença de uma concentração muito baixa de agente redutor (25 mM de DTT), e analisou- se o estado oligomérico do receptor por SDS-PAGE sob condições não redutoras. O R3Mab acelerou significativamente o aparecimento de uma banda de ~ 25 kDa representando o FGFR3S249C monomérico às custas do dímero de ~ 50 kDa, em comparação com os controles Mock ou anticorpo controle (Figura 12C), na verdade, em 2 hr a diminuição dos dímeros foi substancialmente completa na presença de R3Mab. Estes resultados indicam que R3Mab desloca o equilíbrio entre os estados monoméricos e diméricos do FGFR3S249C as custas do monômero.R3MAB NÃO PROMOVE A REGULAÇÃO NEGATIVA DE FGFR3
[558] Os inventores examinaram o efeito do R3Mab (clone 184.6.1) e anticorpos de hibridoma anti-FGFR3 sobre a regulação negativa (downregulation) do FGFR3 analisando a internalização e degradação de FGFR3 em células tratadas com anticorpos FGFR3. As linhagens de células de câncer de bexiga expressando FGFR3 tipo selvagem (RT112) ou FGFR3 mutante (S249C em TCC97 -7) foram tratadas com anticorpos R3Mab ou de hibridoma 1G6 ou 6G1 por 4 a 24 horas, em seguida, as células foram lisadas e os lisados celulares foram coletados para análise dos níveis totais de FGFR3 por Western blot. O tratamento com R3Mab não reduziu os níveis de FGFR3, enquanto o tratamento com mAbs de hibridoma 1G6 e 6G1 reduziu significativamente os níveis de FGFR3. Estes resultados sugerem que R3Mab não promove a regulação negativa de FGFR3, enquanto os mAbs 1G6 e 6G1 promovem a internalização e regulação negativa do receptor FGFR3. Em um experimento separado, os níveis de FGFR3 na superfície foram examinados mediante a análise por FACS. Após 24 horas de tratamento com R3Mab (clone 184.6.1) as células UMUC -14 (contendo a mutação S249C do FGFR3), os níveis de FGFR3 na superfície celular aumentaram ligeiramente. Estes resultados demonstram que o tratamento com R3Mab não promoveu a regulação negativa de FGFR3.R3MAB INIBE O CRESCIMENTO E SINALIZAÇÃO DE FGFR3 EM MODELOS DE TUMORES MÚLTIPLOS
[559] Em seguida, os inventores examinaram o efeito do R3Mab sobre o crescimento in vivo de células de câncer de bexiga. Ele injetou células RT112 (que expressam FGFR3 wt) em camundongos nu/nu, permitindo que os tumores crescessem a um volume médio de ~ 150 mm3, e administraram o veículo ou R3Mab aos animais duas vezes por semana. Comparado com o controle veículo no dia 27, o tratamento com R3Mab a 5 ou 50 mg/kg suprimiu o crescimento do tumor em cerca de 41% ou 73%, respectivamente (Figura 13A). A análise dos lisados tumorais coletados 48 hr ou 72 hr após o tratamento mostraram que o R3Mab diminui acentuadamente o nível de FRS2α fosforilada (Figura 13B). Curiosamente, os níveis de proteína FRS2α total também foram menores nos animais com tumores tratados com R3Mab, sugerindo que a inibição de FGFR3 pode ainda levar a regulação negativa de FRS2α. O R3Mab também reduziu a quantidade de MAPK fosforilada nos tumores, sem afetar os níveis totais de MAPK (Figura 13B). Desse modo, o R3Mab inibe o crescimento de tumores de células tumor RT112 xenoenxertados em conjunto com o bloqueio da sinalização pelo FGFR3 WT.
[560] A seguir, foi investigado o efeito do R3Mab sobre o crescimento de xenoenxertos expressando FGFR3 mutante. O tratamento com R3Mab atenuou profundamente a progressão dos tumores Ba/F3-FGFR3S249C (Figura 13C). Além disso, o R3Mab inibiu significativamente o crescimento de xenoenxertos de carcinoma da bexiga UMUC -14 (Figura 13D). Para avaliar se R3Mab influencia a ativação in vivo de FGFR3S249C, foi avaliado o nível de dímero FGFR3S249C em lisados tumorais coletados 24 horas ou 72 horas após o tratamento. Sob condições não redutoras, a quantidade de dímero FGFR3S249C foi substancialmente mais baixa nos tumores tratados com R3Mab em comparação com grupo controle, enquanto que os níveis totais de FGFR3S249C, a julgar pela quantidade detectada sob condições redutoras, mostrou pouca alteração (Figura 13E). Não foi observado acúmulo aparente do monômero FGFR3S249C nos lisados tumorais, em contraste com os resultados em cultura de células (Figura 13E vs 12A). Isto poderia ser devido à fraca sensibilidade de detecção do FGFR3 monomérico sob condições não redutoras pelo anticorpo policlonal de coelho anti-FGFR3 usado neste estudo (Figura 21). De modo importante, o R3Mab também inibiu significativamente a fosforilação e ativação de MAPK em tumores UMUC-14 (Figura 13E), sugerindo que o R3Mab inibe a atividade in vivo do FGFR3S249C. Os inventores não observaram perda significativa de peso ou outras anormalidades macroscópicas em qualquer um dos estudos in vivo. Além disso, em um estudo de segurança realizado em camundongos, R3Mab, que se liga com afinidade semelhante ao FGFR3 humano e murino, não exerceu qualquer toxicidade discernível em quaisquer órgãos, incluindo a bexiga (dados não mostrados). Juntos, esses dados indicam que exposições múltiplas ao R3Mab são bem toleradas em camundongos.ATIVIDADE ANTI-TUMORAL DO R3MAB EM MODELOS DE XENOENXERTO DE MIELOMA MÚLTIPLO ENVOLVE A ADCC
[561] Para avaliar se o R3Mab poderia abrigar potencial terapêutico para o mieloma múltiplo, os inventores testaram primeiramente o efeito do R3Mab sobre a proliferação e a sobrevida de três linhagens de células t(4; 14)+ em cultura. As células UTMC -2 carregam FGFR3 WT, enquanto a OPM2 e KMS11 carregam uma substituição K650E e Y373C, respectivamente (7). Em cultura, o R3Mab anulou totalmente a proliferação induzida por FGF9 nas células UTMC -2 ( Figura 22A). O R3Mab inibiu modestamente ocrescimento de células OPM2, mas não teve efeito evidente sobre a proliferação de células KMS11 (Figura 22 B, C). Um vez que as células UTMC-2 não formam tumores em camundongos, foi avaliada a eficácia do R3Mab apenas contra os tumores OPM2 e KMS11. O R3Mab aboliu quase que completamente o crescimento do tumor xenoenxertado de ambas as linhagens de células (Figura 14 A, B).
[562] A diferença marcante na atividade de R3Mab contra as células tumorais OPM2 e KMS11 in vitro e in vivo sugeriram a possibilidade de que R3Mab pode ser capaz de suportar funções imunoefetoras mediadas por Fc contra estes tumores superexpressando FGFR3. Ambas as linhas de células expressam altos níveis de CD55 e CD59 (dados não mostrados), dois inibidores da via do complemento e, consequentemente, não foi observada citotoxicidade dependente de complemento (dados não mostrados). Então, os inventores se focaram na ADCC. O ADCC ocorre quando um anticorpo se liga ao seu antígeno sobre uma célula-alvo, e através de sua região Fc, atua nos receptores FCY (FCYRS) expressos nas células imunoefetoras (38). Para avaliar a ADCC in vitro, os inventores incubaram células KMS11 ou OPM2 com células mononucleares do sangue periférico (PBMC) recém isoladas na presença do R3Mab ou anticorpo controle. O R3Mab mediou uma atividade citolítica das PBMC significativa contra ambas linhagens de células de mieloma (Figura 14 C, D). Em contrapartida, o R3Mab não apoiou a citólise de células de câncer de bexiga RT112 ou UMUC-14 (Figura 14 E, F). Conforme mensurado pela análise de Scatchard, as células de mieloma múltiplo expressam substancialmente mais FGFR3 na superfície celular do que as linhagens de células de carcinoma de bexiga (~ 5-6 vezes mais receptores por célula; Figura 23 A, B).
[563] Para abordar a contribuição da ADCC sobre a atividade de R3Mab in vivo, foi introduzido a mutação anteriormente caracterizada D265A/N297A (DANA) no domínio Fc do anticorpo. Esta substituição dupla no domínio Fc de um anticorpo abole a sua ligação ao FCYRS (39), impedindo o recrutamento de células imunoefetoras. A mutação DANA não alterou a ligação de R3Mab ao FGFR3 ou a inibição da atividade de FGFR3 in vitro, nem alterou a farmacocinética do R3Mab em camundongos (dados não mostrados), no entanto, aboliu substancialmente a atividade in vivo contra xenoenxertos OPM2 ou KMS11 (Figura 14 G, H). Em contrapartida, a mutação DANA não alterou a atividade antitumoral do R3Mab para os xenoenxertos de câncer de bexiga RT112 e UMUC -14 (Figura 24 A, B).
[564] Juntos, esses resultados sugerem que a ADCC dependente de Fc desempenha um papel importante na eficácia do R3Mab contra os xenoenxertos mieloma múltiplo OPM2 e KMS11.ESTUDOS ADICIONAIS COM XENOENXERTO
[565] O R3Mab (clone 184.6.1N54S) foi adicionalmente caracterizado da seguinte forma:(a) a eficácia in vivo do R3Mab foi testada utilizando um modelo de tumor xenoenxertado baseado em uma linhagem de células de câncer de fígado (Huh7) essencialmente como descrito anteriormente. Quando testado em uma concentração de anticorpos de 5 mg/kg e 30 mg/kg, o R3Mab inibiu significativamente o crescimento tumoral in vivo. O crescimento tumoral foi inibido a cerca de 50% em relação ao crescimento tumoral nos animais controle.(b) a eficácia in vivo do R3Mab foi testada utilizando um modelo de tumor xenoenxertado baseado em uma linhagem de células de câncer de mama (Cal-51) que expressa FGFR3, essencialmente como descrito anteriormente. Os resultados deste estudo mostraram que a eficácia do anticorpo R3Mab foi capaz de inibir os tumores in vivo quando testados em concentrações de anticorpos na escala de 1 mg/kg a 100 mg/kg. O crescimento tumoral foi inibido a cerca de 30% em relação ao crescimento tumoral nos animais controle.DISCUSSÃO
[566] A associação da superexpressão de FGFR3 com mau prognóstico em pacientes com mieloma múltiplo t(4; 14)+ e a atividade transformadora do FGFR3 ativado em diversos modelos experimentais estabeleceu o FGFR3 como um fator oncogênico importante e, portanto, um alvo terapêutico potencial nessa malignidade hematológica. Por outro lado, apesar dos relatos de uma alta frequência de mutação e/ou superexpressão de FGFR3 no carcinoma de bexiga (24, 25, 40), um papel fundamental para a sinalização de FGFR3 nesta malignidade epitelial não foi estabelecido em estudos in vivo. Além disso, o potencial terapêutico da inibição de FGFR3 no câncer de bexiga ainda não foi definido. Na presente invenção os inventores demonstraram que a intervenção genética ou farmacológica com FGFR3 inibe o crescimento de diversos xenoenxertos de cânceres de bexiga humanos em camundongos. Estes resultados demonstram que a função de FGFR3 é importante para o crescimento tumoral neste ambiente, ressaltando a importância potencial deste receptor como um fator oncogênico e alvo terapêutico no câncer de bexiga. Bloqueio da função FGFR3 inibiu o crescimento de xenoenxertos expressando FGFR3 tanto WT quanto mutante similares, sugerindo que ambas as formas de receptor podem contribuir significativamente para a progressão tumoral na bexiga. Embora com uma frequência muito menor do que em câncer de bexiga, as mutações ou superexpressão de FGFR3 foram identificadas em outras neoplasias malignas de tumores sólidos, incluindo o carcinoma cervical (40), carcinoma hepatocelular (41) e câncer de pulmão não pequenas células (42, 43), sugerindo uma potencial contribuição do FGFR3 em outros tipos de cânceres epiteliais.
[567] O aparente envolvimento do FGFR3 em diversas neoplasias malignas identifica este receptor como um candidato interessante para uma terapia direcionada. Enquanto compostos de pequenas moléculas que podem inibir a atividade quinase do FGFR3 foram descritas (18-22, 44), a homologia próxima dos domínios quinase na família FGFR tem dificultado o desenvolvimento de inibidores seletivos para FGFR3. A falta de seletividade dos inibidores anteriormente descritos torna difícil discernir a contribuição relativa do FGFR3 para a biologia de cânceres de tipos específicos; além disso, pode carregar obrigações de segurança, limitando os níveis de dose máxima e, assim, limitando a inibição ideal de FGFR3. Portanto, para atingir metas seletivas e específicas ao FGFR3, os inventores se voltaram para uma estratégia baseada em anticorpos. Eles avaliaram que um anticorpo terapêutico ideal deve ser capaz de bloquear não só o FGFR3 tipo selvagem, como também os mutantes de FGFR3 prevalentes ligados ao câncer. Além disso, dado que a dimerização do FGFR3 é fundamental para sua ativação, um anticorpo que não apenas bloqueia o ligante como também interfere com a dimerização do receptor poderia ser superior. Dentre as propriedades adicionais desejáveis, pode-se incluir a capacidade de suportar a função efetora mediada por Fc e uma maior meia vida no soro conferida pela região estrutural natural de um anticorpo de comprimento total. Os inventores se concentraram nos esforços de triar e desenvolver uma molécula de anticorpo que combinasse todas essas características, levando à geração do R3Mab. Estudos de ligação demonstraram a capacidade do R3Mab em competir com os ligantes FGF pela interação com ambas isoformas de FGFR3, IIIb e IIIc. Outros experimentos com linhagens de células BAF/3 transfectadas confirmaram a notável capacidade do R3Mab em bloquear tanto o FGFR3 WT quanto os mutantes de FGFR3 prevalentes associados ao câncer. Além disso, o R3Mab exerceu uma atividade antitumoral significativa em diversos modelos de xenoenxerto de câncer de bexiga expressando tanto FGFR3 WT quanto FGFR3S249C, que é o a forma mutante mais comum do receptor nesta doença. Os estudos farmacodinâmicos sugerem que a atividade antitumoral do R3Mab nesses modelos é baseada na inibição da sinalização de FGFR3, evidente pela fosforilação diminuída de seus mediadores a jusante, FRS2α e MAPK. Estes dados reforçam a conclusão de que o FGFR3 é necessário para a progressão do tumor de bexiga, conforme demonstrado pelos nossos estudos com shRNA para FGFR3.
[568] As mutações de FGFR3 no câncer de bexiga representam uma das alterações oncogênicas mais frequentes de uma proteína quinase nas neoplasias malignas de tumores sólidos, que lembra a mutação comum de B- Raf no melanoma (45). A maioria das mutações ativadoras no FGFR3 dá origem a uma cisteína não pareada, levando a dimerização do receptor independente de ligante e vários graus de ativação constitutiva. Um estudo anterior, usando um fragmento Fab anti-FGFR3 monovalente indicou atividade inibitória diferencial contra mutantes FGFR3 específicos (46), no entanto, a base molecular para este efeito variável não foi investigada. Em comparação com fragmentos de anticorpos monovalentes, anticorpos bivalentes têm a capacidade de induzir o agrupamento de antígenos, e no caso de receptor tirosina quinase, podem causar oligomerização e ativação do receptor. Apesar de seu comprimento total, configuração bivalente, o R3Mab exibiu inibição universal do FGFR3 WT e de um amplo espectro de FGFR3 mutantes, incluindo as variantes que são dependentes de ligante (FGFR3G372C, FGFR3Y375C), constitutivamente ativos (FGFR3R248C, FGFR3S249C), ou ambos (FGFR3K652E). Estes resultados levantam a questão: Como é que o R3Mab antagoniza tanto o FGFR3 WT quanto os diversos mutantes de FGFR3, incluindo as variantes ligadas por dissulfeto?
[569] Com base no alinhamento de sequências com FGFR1, o peptídeo epítopo reconhecido por R3Mab se sobrepõe com os resíduos de FGFR3 envolvidos na ligação com o ligante, com a heparina, bem como com a dimerização do receptor. Esta conclusão foi confirmada por estudos de cristalografia do complexo entre R3Mab e as regiões extracelulares do FGFR3. A estrutura de raios-X revelou que o anticorpo se liga a regiões de IgD2 e IgD3 que são fundamentais para a interação ligante-receptor, assim como para o contato receptor-receptor. Portanto, R3Mab pode bloquear o FGFR3 WT tanto pela competição com os ligantes quanto pela prevenção da dimerização do receptor. O R3Mab pode empregar um mecanismo semelhante para inibir FGFR3K652E, que tem baixa atividade constitutiva, mas necessita do ligante para a ativação completa. Além disso, a ligação do R3Mab modifica a orientação relativa do IgD3 do FGFR3 com relação ao IgD2. Esta revelação levanta a possibilidade formal de que o anticorpo pode também inibir a ativação do receptor, forçando uma conformação que não é propícia para a transdução de sinal - uma noção que requer um estudo mais aprofundado.
[570] Para obter uma melhor visão sobre como R3Mab bloqueia as variantes de FGFR3 que possuem uma cisteína não pareada, os inventores analisaram mais detalhadamente o mutante mais comum, FGFR3S249C. Os experimentos com o bloqueador sem sulfidrila DTNB indicaram um equilíbrio dinâmico entre o estado monomérico e dimérico do FGFR3S249C. Equilíbrio semelhante entre os estados oxidado e reduzido modulados por reguladores redox endógenos foram relatados para os receptores NMDA (46). A incubação de células de câncer de bexiga expressando FGFR3S249C com R3Mab levaram a um declínio na quantidade de receptores dímeros e um concomitante aumento no nível de monômeros. Além disso, o fragmento purificado IgD2-D3 de FGFR3S249C dímeros formados em solução, e quando incubado com R3Mab, os dímeros de forma constante desaparecido enquanto FGFR3S249C monomérica acumulado. Tomados em conjunto com a análise estrutural, estes resultados sugerem que o R3Mab captura o FGFR3S249C monomérico e dificulta a sua dimerização. Com o tempo, R3Mab desloca o equilíbrio para o estado monomérico, bloqueando a atividade do receptor constitutivo. Este mecanismo também poderia explicar como o R3Mab inibe outros mutantes de cisteína do FGFR3.
[571] Outro achado importante deste estudo foi a potente atividade anti-tumoral in vivo do R3Mab contra diversas linhagens de células de mieloma t (4; 14)+; OPM2 e KMS11. Em contraste, o R3Mab teve um impacto modesto ou mínimo sobre a proliferação ou sobrevivência dessas células em cultura. As células OPM2 e KMS11 expressam níveis relativamente altos de FGFR3 na superfície celular (5-6 vezes mais do que as células de carcinoma de bexiga RT112 e UMUC-14). Estas densidades mais elevadas de antígeno podem permitir que o R3Mab apoie o recrutamento eficiente de células imunoefetoras carregando FCYR e a ativação da ADCC. De fato, na presença de PBMCs humanas, o R3Mab medeia a citólise de células OPM2 e KMS11, mas não de células cancerosas de bexiga RT112 ou UMUC -14. Além disso, a versão mutante DANA do R3Mab, que é incapaz de se ligar ao FCYR, não teve efeito sobre o crescimento in vivo de células OPM2 ou KMS11, mas ainda assim suprimiu o crescimento de tumores RT112 e UMUC-14 de forma semelhante ao R3Mab. Em conjunto, esses dados indicam que o R3Mab possui um duplo mecanismo de atividade antitumoral. (a) Nas células que expressam níveis mais baixos de FGFR3 WT ou mutante na superfície, ele bloqueia a sinalização dependente de ligante ou constitutiva, (b) Nas células que expressam níveis de FGFR3 relativamente altos na superfície, ele induz a ADCC.
[572] Nossos resultados também levantam algumas novas questões. Em primeiro lugar, não se sabe por que as linhagens de células de câncer de bexiga testadas neste estudo exibiram sensibilidade variável ao R3Mab. Tal resposta diferencial, que é comum para terapia alvo, pode ser um reflexo da composição genética distinta dos tumores individuais. De fato, células de câncer de mama HER2-positivas mostraram sensibilidade variável para anticorpos anti-Her2 (48), assim como as diversas células de câncer em resposta ao anticorpo anti-EGFR (49). Neste contexto, o desenvolvimento de novos modelos in vivo de câncer de bexiga com FGFR3 WT e mutante é urgentemente necessário para avaliar a sensibilidade das moléculas FGFR3 em animais. Além disso, a elucidação de biomarcadores preditivos pode ajudar a identificar pacientes que podem se beneficiar de forma otimizada com a terapia direcionada para FGFR3. Em segundo lugar, pelo fato do R3Mab não induzir a regressão do tumor nos modelos que foram examinados, estudos futuros devem explorar se o R3Mab pode cooperar com agentes terapêuticos estabelecidos.
[573] Em conclusão, nossos resultados demonstram o envolvimento do FGFR3, tanto WT como mutante, como importante para o crescimento do câncer de bexiga, expandindo a participação oncogênica in vivo desse receptor das malignidades hematológicas para as epiteliais. Além disso, nossos resultados demonstram que o FGFR3 tanto WT como mutante pode ser eficazmente direcionada em tumores com um anticorpo de comprimento total que combina a capacidade de bloquear a ligação do ligante, a dimerização e sinalização do receptor, bem como promover a lise de células tumorais pela ADCC. Estes resultados fornecem um forte argumento para a investigação baseada em anticorpos, terapias que têm o FGFR3 como alvo em diversas malignidades associadas a este receptor.LISTA DE REFERÊNCIA PARCIAL1. Eswarakumar, V.P., Lax, I., e Schlessinger, J. 2005. Cellularsignaling by fibroblast growth factor receptors. Cytokine Growth Factor Rev 16:139-149.2. L'Hote, C.G., e Knowles, M.A. 2005. Cell responses toFGFR3 signalling: growth, differentiation and apoptosis. Exp Cell Res 304:417431.3. Dailey, L., Ambrosetti, D., Mansukhani, A., e Basilico, C.2005. Mechanisms underlying differential responses to FGF signaling. Cytokine Growth Factor Rev 16:233-247.4. Mohammadi, M., Olsen, S.K., e Ibrahimi, O.A. 2005.Structural basis for fibroblast growth factor receptor activation. Cytokine Growth Factor Rev 16:107-137.5. Grose, R., e Dickson, C. 2005. Fibroblast growth factorsignaling in tumorigenesis. Cytokine Growth Factor Rev 16:179-186.6. Chang, H., Stewart, A.K., Qi, X.Y., Li, Z.H., Yi, Q.L., e Trudel,S. 2005. Immunohistochemistry accurately predicts FGFR3 aberrant expression and t(4;14) in multiple myeloma. Blood 106:353-355.7. Chesi, M., Nardini, E., Brents, L.A., Schrock, E., Ried, T.,Kuehl, W.M., e Bergsagel, P.L. 1997. Frequent translocation t(4;14)(p16.3;q32.3) in multiple myeloma is associated with increased expression and activating mutations of fibroblast growth factor receptor 3. Nat Genet 16:260-264.8. Fonseca, R., Blood, E., Rue, M., Harrington, D., Oken, M.M.,Kyle, R.A., Dewald, G.W., Van Ness, B., Van Wier, S.A., Henderson, K.J., et al.. 2003. Clinical and biologic implications of recurrent genomic aberrations in myeloma. Blood 101:4569-4575.9. Moreau, P., Facon, T., Leleu, X., Morineau, N., Huyghe, P.,Harousseau, J.L., Bataille, R., e Avet-Loiseau, H. 2002. Recurrent 14q32 translocations determine the prognosis of multiple myeloma, especially in patients receiving intensive chemotherapy. Blood 100:1579-1583.10. Pollett, J.B., Trudel, S., Stern, D., Li, Z.H., e Stewart, A.K. 2002. Overexpression of the myeloma-associated oncogene fibroblast growth factor receptor 3 confers dexamethasone resistance. Blood 100:3819-3821.11. Bernard-Pierrot, I., Brams, A., Dunois-Larde, C., Caillault, A., Diez de Medina, S.G., Cappellen, D., Graff, G., Thiery, J.P., Chopin, D., Ricol, D., et al.. 2006. Oncogenic properties of the mutated forms of fibroblast growth factor receptor 3b. Carcinogenesis 27:740-747.12. Agazie, Y.M., Movilla, N., Ischenko, I., e Hayman, M.J. 2003. The phosphotyrosine phosphatase SHP2 is a critical mediator of transformation induced by the oncogenic fibroblast growth factor receptor 3. Oncogene 22:69096918.13. Ronchetti, D., Greco, A., Compasso, S., Colombo, G., Dell'Era, P., Otsuki, T., Lombardi, L., e Neri, A. 2001. Deregulated FGFR3 mutants in multiple myeloma cell lines with t(4;14): comparative analysis of Y373C, K650E and the novel G384D mutations. Oncogene 20:3553-3562.14. Chesi, M., Brents, L.A., Ely, S.A., Bais, C., Robbiani, D.F., Mesri, E.A., Kuehl, W.M., e Bergsagel, P.L. 2001. Activated fibroblast growth factor receptor 3 is an oncogene that contributes to tumor progression in multiple myeloma. Blood 97:729-736. 15. Plowright, E.E., Li, Z., Bergsagel, P.L., Chesi, M., Barber, D.L., Branch, D.R., Hawley, R.G., e Stewart, A.K. 2000. Ectopic expression of fibroblast growth factor receptor 3 promotes myeloma cell proliferation and prevents apoptosis. Blood 95:992-998.16. Chen, J., Williams, I.R., Lee, B.H., Duclos, N., Huntly, B.J., Donoghue, D.J., e Gilliland, D.G. 2005. Constitutively activated FGFR3 mutants signal through PLCgamma-dependent and -independent pathways for hematopoietic transformation. Blood 106:328-337.17. Li, Z., Zhu, Y.X., Plowright, E.E., Bergsagel, P.L., Chesi, M., Patterson, B., Hawley, T.S., Hawley, R.G., e Stewart, A.K. 2001. The myeloma- associated oncogene fibroblast growth factor receptor 3 is transforming in hematopoietic cells. Blood 97:2413-2419.18. Trudel, S., Ely, S., Farooqi, Y., Affer, M., Robbiani, D.F., Chesi, M., e Bergsagel, P.L. 2004. Inhibition of fibroblast growth factor receptor 3 induces differentiation and apoptosis in t(4;14) myeloma. Blood 103:3521-3528.19. Trudel, S., Li, Z.H., Wei, E., Wiesmann, M., Chang, H., Chen, C., Reece, D., Heise, C., e Stewart, A.K. 2005. CHIR-258, a novel, multitargeted tyrosine kinase inhibitor for the potential treatment of t(4;14) multiple myeloma. Blood 105:2941-2948.20. Chen, J., Lee, B.H., Williams, I.R., Kutok, J.L., Mitsiades, C.S., Duclos, N., Cohen, S., Adelsperger, J., Okabe, R., Coburn, A., et al. 2005. FGFR3 as a therapeutic target of the small molecule inhibitor PKC412 in hematopoietic malignancies. Oncogene 24:8259-8267.21. Paterson, J.L., Li, Z., Wen, X.Y., Masih-Khan, E., Chang, H., Pollett, J.B., Trudel, S., e Stewart, A.K. 2004. Preclinical studies of fibroblast growth factor receptor 3 as a therapeutic target in multiple myeloma. Br J Haematol 124:595-603.22. Grand, E.K., Chase, A.J., Heath, C., Rahemtulla, A., e Cross, N.C. 2004. Targeting FGFR3 in multiple myeloma: inhibition of t(4;14)-positive cells by SU5402 and PD173074. Leukemia 18:962-966.23. Gomez-Roman, J.J., Saenz, P., Molina, M., Cuevas Gonzalez, J., Escuredo, K., Santa Cruz, S., Junquera, C., Simon, L., Martinez, A., Gutierrez Banos, J.L., et al. 2005. Fibroblast growth factor receptor 3 is overexpressed in urinary tract carcinomas and modulates the neoplastic cell growth. Clin Cancer Res 11:459-465.24. Tomlinson, D.C., Baldo, O., Harnden, P., e Knowles, M.A. 2007. FGFR3 protein expression and its relationship to mutation status and prognostic variables in bladder cancer. J Pathol 213:91-98.25. van Rhijn, B.W., Montironi, R., Zwarthoff, E.C., Jobsis, A.C., e van der Kwast, T.H. 2002. Frequent FGFR3 mutations in urothelial papilloma. J Pathol 198:245-251.26. Tomlinson, D.C., Hurst, C.D., e Knowles, M.A. 2007. Knockdown by shRNA identifies S249C mutant FGFR3 as a potential therapeutic target in bladder cancer. Oncogene 26:5889-5899.27. Martinez-Torrecuadrada, J., Cifuentes, G., Lopez-Serra, P., Saenz, P., Martinez, A., e Casal, J.I. 2005. Targeting the extracellular domain of fibroblast growth factor receptor 3 with human single-chain Fv antibodies inhibits bladder carcinoma cell line proliferation. Clin Cancer Res 11:6280-6290.28. Martinez-Torrecuadrada, J.L., Cheung, L.H., Lopez-Serra, P., Barderas, R., Canamero, M., Ferreiro, S., Rosenblum, M.G., e Casal, J.I. 2008. Antitumor activity of fibroblast growth factor receptor 3-specific immunotoxins in a xenograft mouse model of bladder carcinoma is mediated by apoptosis. Mol Cancer Ther 7:862-873.29. Ornitz, D.M., e Leder, P. 1992. Ligand specificity and heparin dependence of fibroblast growth factor receptors 1 and 3. J Biol Chem 267:16305-16311. 30. d'Avis, P.Y., Robertson, S.C., Meyer, A.N., Bardwell, W.M., Webster, M.K., e Donoghue, D.J. 1998. Constitutive activation of fibroblast growth factor receptor 3 by mutations responsible for the lethal skeletal dysplasia thanatophoric dysplasia type I. Cell Growth Differ 9:71-78.31. Adar, R., Monsonego-Ornan, E., David, P., e Yayon, A. 2002. Differential activation of cysteine-substitution mutants of fibroblast growth factor receptor 3 is determined by cysteine localization. J Bone Miner Res 17:860-868.32. Knowles, M.A. 2008. Novel therapeutic targets in bladder cancer: mutation and expression of FGF receptors. Future Oncol 4:71-83.33. Naski, M.C., Wang, Q., Xu, J., e Ornitz, D.M. 1996. Graded activation of fibroblast growth factor receptor 3 by mutations causing achondroplasia and thanatophoric dysplasia. Nat Genet 13:233-237.34. Plotnikov, A.N., Schlessinger, J., Hubbard, S.R., e Mohammadi, M. 1999. Structural basis for FGF receptor dimerization and activation. Cell 98:641-650.35. Olsen, S.K., Ibrahimi, O.A., Raucci, A., Zhang, F., Eliseenkova, A.V., Yayon, A., Basilico, C., Linhardt, R.J., Schlessinger, J., e Mohammadi, M. 2004. Insights into the molecular basis for fibroblast growth factor receptor autoinhibition and ligand-binding promiscuity. Proc Natl Acad Sci U S A 101:935-940.36. Jebar, A.H., Hurst, C.D., Tomlinson, D.C., Johnston, C., Taylor, C.F., e Knowles, M.A. 2005. FGFR3 and Ras gene mutations are mutually exclusive genetic events in urothelial cell carcinoma. Oncogene 24:5218-5225.37. Ellman, G.L. 1959. Tissue sulfhydryl groups. Arch Biochem Biophys 82:70-77.38. Adams, G.P., e Weiner, L.M. 2005. Monoclonal antibody therapy of cancer. Nat Biotechnol 23:1147-1157.39. Gong, Q., Ou, Q., Ye, S., Lee, W.P., Cornelius, J., Diehl, L., Lin, W.Y., Hu, Z., Lu, Y., Chen, Y., et al. 2005. Importance of cellular microenvironment and circulatory dynamics in B cell immunotherapy. J Immunol 174:817-826.40. Cappellen, D., De Oliveira, C., Ricol, D., de Medina, S., Bourdin, J., Sastre-Garau, X., Chopin, D., Thiery, J.P., e Radvanyi, F. 1999. Frequent activating mutations of FGFR3 in human bladder and cervix carcinomas. Nat Genet 23:18-20.41. Qiu, W.H., Zhou, B.S., Chu, P.G., Chen, W.G., Chung, C., Shih, J., Hwu, P., Yeh, C., Lopez, R., e Yen, Y. 2005. Over-expression of fibroblast growth factor receptor 3 in human hepatocellular carcinoma. WOrld J Gastroenterol 11:5266-5272.42. Cortese, R., Hartmann, O., Berlin, K., e Eckhardt, F. 2008. Correlative gene expression and DNA methylation profiling in lung development nominate new biomarkers in lung cancer. Int J Biochem Cell Biol 40:1494-1508.43. WOenckhaus, M., Klein-Hitpass, L., Grepmeier, U., Merk, J., Pfeifer, M., Wild, P., Bettstetter, M., Wuensch, P., Blaszyk, H., Hartmann, A., et al. 2006. Smoking and cancer-related gene expression in bronchial epithelium and non-small-cell lung cancers. J Pathol 210:192-204.44. Xin, X., Abrams, T.J., Hollenbach, P.W., Rendahl, K.G., Tang, Y., Oei, Y.A., Embry, M.G., Swinarski, D.E., Garrett, E.N., Pryer, N.K., et al. 2006. CHIR-258 is efficacious in a newly developed fibroblast growth factor receptor 3-expressing orthotopic multiple myeloma model in mice. Clin Cancer Res 12:4908-4915.45. Davies, H., Bignell, G.R., Cox, C., Stephens, P., Edkins, S., Clegg, S., Teague, J., WOffendin, H., Garnett, M.J., Bottomley, W., et al. 2002. Mutations of the BRAF gene in human cancer. Nature 417:949-954.46. Trudel, S., Stewart, A.K., Rom, E., Wei, E., Li, Z.H., Kotzer, S., Chumakov, I., Singer, Y., Chang, H., Liang, S.B., et al. 2006. The inhibitory anti-FGFR3 antibody, PRO-001, is cytotoxic to t(4;14) multiple myeloma cells. Blood 107:4039-4046.47. Gozlan, H., e Ben-Ari, Y. 1995. NMDA receptor redox sites: are they targets for selective neuronal protection? Trends Pharmacol Sci 16:368-374.48. Hudziak, R.M., Lewis, G.D., Winget, M., Fendly, B.M., Shepard, H.M., e Ullrich, A. 1989. p185HER2 monoclonal antibody has antiproliferative effects in vitro and sensitizes human breast tumor cells to tumor necrosis factor. Mol Cell Biol 9:1165-1172.49. Masui, H., Kawamoto, T., Sato, J.D., WOlf, B., Sato, G., e Mendelsohn, J. 1984. Growth inhibition of human tumor cells in athymic mice by anti-epidermal growth factor receptor monoclonal antibodies. Cancer Res 44:1002-1007.50. Pai, R., Dunlap, D., Qing, J., Mohtashemi, I., Hotzel, K., e French, D.M. 2008. Inhibition of fibroblast growth factor 19 reduces tumor growth by modulating beta-catenin signaling. Cancer Res 68:5086-5095.51. Pegram, M., Hsu, S., Lewis, G., Pietras, R., Beryt, M., Sliwkowski, M., Coombs, D., Baly, D., Kabbinavar, F., e Slamon, D. 1999. Inhibitory effects of combinations of HER-2/neu antibody and chemotherapeutic agents used for treatment of human breast cancers. Oncogene 18:2241-2251.52. Lee, C.V., Liang, W.C., Dennis, M.S., Eigenbrot, C., Sidhu, S.S., e Fuh, G. 2004. High-affinity human antibodies from phage-displayed synthetic Fab libraries with a single frameWOrk scaffold. J Mol Biol 340:10731093.53. Liang, W.C., Dennis, M.S., Stawicki, S., Chanthery, Y., Pan, Q., Chen, Y., Eigenbrot, C., Yin, J., Koch, A.W., Wu, X., et al. 2007. Function blocking antibodies to neuropilin-1 generated from a designed human synthetic antibody phage library. J Mol Biol 366:815-829.54. Carter, P., Presta, L., Gorman, C.M., Ridgway, J.B., Henner, D., WOng, W.L., Rowland, A.M., Kotts, C., Carver, M.E., e Shepard, H.M. 1992. Humanization of an anti-p185HER2 antibody for human cancer therapy. Proc Natl Acad Sci U S A 89:4285-4289.55. Sidhu, S.S., Li, B., Chen, Y., Fellouse, F.A., Eigenbrot, C., e Fuh, G. 2004. Phage-displayed antibody libraries of synthetic heavy chain complementarity determining regions. J Mol Biol 338:299-310.56. Otwinowski, Z.a.M., W. 1997. Processing of X-ray diffraction data collected in oscillation mode. Methods in Enzymology 276:307326.57. McCoy, A.J., Grosse-Kunstleve, R.W., Storoni, L.C., e Read, R.J. 2005. Likelihood-enhanced fast translation functions. Acta Crystallogr D Biol Crystallogr 61:458-464.58. Emsley, P., e Cowtan, K. 2004. Coot: model-building tools for molecular graphics. Acta Crystallogr D Biol Crystallogr 60:2126-2132.59. Murshudov, G.N., Vagin, A.A., e Dodson, E.J. 1997. Refinement of macromolecular structures by the maximum-likelihood method. Acta Crystallogr D Biol Crystallogr 53:240-255.574. ] Embora a presente invenção acima divulgada tenha sido descrita em detalhes por meio de ilustrações e exemplos para fins de clareza de entendimento, as descrições e os exemplos não devem ser interpretados como limitantes do escopo da presente invenção.
Claims (38)
1. ANTICORPO ANTAGONISTA ANTI-FGFR3, caracterizadopor compreender:(a) uma região variável de cadeia leve compreendendo (i) HVR-L1 compreendendo a sequência de aminoácidos de SEQ ID NO:87, (ii) HVR-L2 compreendendo a sequência de aminoácidos de SEQ ID NO:88, (iii) HVR-L3 compreendendo a sequência de aminoácidos de SEQ ID NO:89, e(b) uma região variável de cadeia pesada compreendendo (i) HVR- H1 compreendendo a sequência de aminoácidos de SEQ ID NO:84, (ii) HVR-H2 compreendendo a sequência de aminoácidos de SEQ ID NO:85, e (iii) HVR-H3 compreendendo a sequência de aminoácidos de SEQ ID NO:86;em que o anticorpo se liga ao FGFR3 humano com um Kd de 1 x 10-8 ou mais forte e em que o anticorpo inibe a dimerização do receptor FGFR3 com outra unidade do receptor, através do qual a ativação do receptor é inibida.
2. ANTICORPO, de acordo com a reivindicação 1,caracterizado por se ligar a FGFR3-IIIb e FGFR3-IIIc.
3. ANTICORPO, de acordo com a reivindicação 1,caracterizado por possuir pouca ou nenhuma função agonista.
4. ANTICORPO, de acordo com a reivindicação 1,caracterizado por possuir função efetora.
5. ANTICORPO, de acordo com a reivindicação 4,caracterizado pela função efetora compreender citotoxicidade mediada por células dependente de anticorpo.
6. ANTICORPO, de acordo com a reivindicação 5,caracterizado por ser capaz de matar uma célula de mieloma múltiplo compreendendo uma translocação t(4;14).
7. ANTICORPO, de acordo com a reivindicação 6,caracterizado por ser um anticorpo nu.
8. ANTICORPO, de acordo com a reivindicação 5,caracterizado por ser capaz de matar uma célula expressando 10.000 ou mais moléculas de FGFR3.
9. ANTICORPO, de acordo com a reivindicação 1,caracterizado por se ligar a um polipeptídeo que compreende uma sequência de aminoácidos LAVPAANTVRFRCPA (SEQ ID NO: 179) ouSDVEFHCKVYSDAQP (SEQ ID NO: 180).
10. ANTICORPO, de acordo com a reivindicação 1,caracterizado por inibir a atividade constitutiva de FGFR3.
11. ANTICORPO, de acordo com a reivindicação 1,caracterizado pela atividade constitutiva de FGFR3 ser ligante-dependente.
12. ANTICORPO, de acordo com a reivindicação 10,caracterizado pela atividade constitutiva de FGFR3 ser ligante-independente.
13. ANTICORPO, de acordo com a reivindicação 1,caracterizado por inibir FGFR3 compreendendo uma mutação correspondente a FGFR3-IIIbR248C.
14. ANTICORPO, de acordo com a reivindicação 1, caracterizado por inibir FGFR3 compreendendo uma mutação correspondente a FGFR3- IIIbK652E.
15. ANTICORPO, de acordo com a reivindicação 1, caracterizado por inibir FGFR3 compreendendo uma mutação correspondente a FGFR3- IIIbS249C.
16. ANTICORPO, de acordo com a reivindicação 1, caracterizado por inibir FGFR3 compreendendo uma mutação correspondente a FGFR3- IIIbG372C.
17. ANTICORPO, de acordo com a reivindicação 1, caracterizado por inibir FGFR3 compreendendo uma mutação correspondente a FGFR3- IIIbY375C.
18. ANTICORPO, de acordo com a reivindicação 1, caracterizado por compreender uma região variável de cadeia pesada que compreende a SEQ ID NO 132 e uma região variável de cadeia leve.
19. ANTICORPO, de acordo com a reivindicação 1, caracterizado por compreender uma região variável de cadeia pesada e uma região variável de cadeia leve que compreende a SEQ ID NO 133.
20. ANTICORPO, de acordo a reivindicação 1, caracterizado por compreender uma região variável de cadeia pesada que compreende a SEQ ID NO 132, e uma região variável de cadeia leve que compreende a SEQ ID NO 133.
21. ANTICORPO, de acordo com a reivindicação 1, caracterizado por pelo menos uma porção da sequência da região estrutural ser uma sequência da região estrutural de consenso humano.
22. ANTICORPO, de acordo com a reivindicação 1, caracterizado por compreender uma sequência de região estrutural de consenso humano subgrupo K.
23. ANTICORPO, de acordo com a reivindicação 1, caracterizado por compreender uma sequência de região estrutural de consenso humano subgrupo III.
24. ANTICORPO, de acordo com a reivindicação 1,caracterizado por ser um anticorpo monoclonal.
25. ANTICORPO, de acordo com a reivindicação 1,caracterizado por ser selecionado a partir do grupo que consiste em um anticorpo quimérico, um anticorpo humanizado, um anticorpo maturado por afinidade, um anticorpo humano e um anticorpo biespecífico.
26. ANTICORPO, de acordo com a reivindicação 1, caracterizado por ser um fragmento de anticorpo.
27. ANTICORPO MONOCLONAL antagonista ANTI-FGFR3, caracterizado por compreender: (a) uma região variável de cadeia leve compreendendo (i) HVR-L1 que compreende a sequência de aminoácidos de SEQ ID NO: 87, (ii) HVR-L2 que compreende a sequência de aminoácidos de SEQ ID NO: 88, (iii) HVR-L3 que compreende a sequência de aminoácidos de SEQ ID NO: 89, e(b) uma região variável de cadeia pesada compreendendo (i) HVR- H1 que compreende a sequência de aminoácidos de SEQ ID NO: 84, (ii) HVR- H2 que compreende a sequência de aminoácidos de SEQ ID NO: 85, e (iii) HVR- H3 que compreende a sequência de aminoácidos de SEQ ID NO: 86,em que o anticorpo se liga ao FGFR3 humano com um Kd de 1 x 10-8 ou mais forte e em que o anticorpo inibe a dimerização do receptor FGFR3 com outra unidade do receptor, através do qual a ativação do receptor é inibida.
28. POLINUCLEOTÍDEO, caracterizado por codificar um anticorpo, conforme definido em qualquer uma das reivindicações 1 a 27.
29. VETOR, caracterizado por compreender o polinucleotídeo, conforme definido na reivindicação 28.
30. MICRO-ORGANISMO TRANSGÊNICO, in vitro,caracterizado por compreender o vetor, conforme definido na reivindicação 29.
31. MÉTODO PARA PRODUÇÃO DE UM ANTICORPO ANTI- FGFR3, caracterizado por compreender cultivar o micro-organismo transgênico, que compreende um polinucleotídeo, que codifica o anticorpo, conforme definido em qualquer uma das reivindicações 1 a 27, de modo que o polinucleotídeo seja expresso e, opcionalmente, recuperar o anticorpo da cultura.
32. FORMULAÇÃO FARMACÊUTICA, caracterizada por compreender o anticorpo, conforme definido em qualquer uma das reivindicações 1 a 27, e um veículo farmaceuticamente aceitável.
33. USO DO ANTICORPO ANTAGONISTA ANTI-FGFR3, conforme definido em qualquer uma das reivindicações 1 a 27, caracterizado por ser na manufatura de um medicamento para inibição da proliferação celular e/ou depleção de células de câncer.
34. USO DO ANTICORPO ANTAGONISTA ANTI-FGFR3, conforme definido em qualquer uma das reivindicações 1 a 27, caracterizado por ser na manufatura de um medicamento para tratar câncer.
35. USO, de acordo com a reivindicação 34, caracterizado pelo câncer ser selecionado a partir do grupo que consiste em: tumor sólido, mieloma múltiplo, carcinoma de bexiga, câncer de pulmão de células não pequenas, câncer de ovário, câncer da tireoide, câncer de cabeça e pescoço, câncer de fígado, carcinoma de mama, câncer gástrico, carcinoma de células de transição e câncer colorretal.
36. USO, de acordo com a reivindicação 34, caracterizado pelo câncer estar associado com a ativação de FGFR3 e/ou superexpressão de FGFR3.
37. USO DO ANTICORPO ANTAGONISTA ANTI-FGFR3, conforme definido em qualquer uma das reivindicações 1 a 27, caracterizado por ser na manufatura de um medicamento para tratar ou prevenir um distúrbio esquelético.
38. USO, de acordo com a reivindicação 37, caracterizado pelo distúrbio esquelético ser selecionado a partir do grupo que consiste em: acondroplasia, hipocondroplasia, nanismo, displasia tanatofórica e síndrome de craniosinostose.
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US16322209P | 2009-03-25 | 2009-03-25 | |
| US61/163,222 | 2009-03-25 | ||
| PCT/US2010/028470 WO2010111367A1 (en) | 2009-03-25 | 2010-03-24 | Anti-fgfr3 antibodies and methods using same |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| BRPI1006448A2 BRPI1006448A2 (pt) | 2020-08-18 |
| BRPI1006448B1 true BRPI1006448B1 (pt) | 2021-08-17 |
Family
ID=42340706
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| BRPI1006448-6A BRPI1006448B1 (pt) | 2009-03-25 | 2010-03-24 | Anticorpo antagonista anti-fgfr3, anticorpo monoclonal, polinucleotídeo, vetor, microorganismo transgênico, método para produção de um anticorpo anti-fgfr3, formulação farmacêutica e usos do anticorpo antagonista anti-fgfr3 |
Country Status (29)
| Country | Link |
|---|---|
| US (10) | US20100291114A1 (pt) |
| EP (3) | EP2411414B1 (pt) |
| JP (1) | JP5757534B2 (pt) |
| KR (2) | KR101822663B1 (pt) |
| CN (2) | CN102378767B (pt) |
| AR (2) | AR075939A1 (pt) |
| AU (1) | AU2010229994B2 (pt) |
| BR (1) | BRPI1006448B1 (pt) |
| CA (1) | CA2754163C (pt) |
| CO (1) | CO6450665A2 (pt) |
| DK (2) | DK3702371T3 (pt) |
| EC (1) | ECSP11011417A (pt) |
| ES (2) | ES2549769T3 (pt) |
| HK (1) | HK1167666A1 (pt) |
| HR (2) | HRP20221471T1 (pt) |
| HU (2) | HUE025726T2 (pt) |
| IL (2) | IL214433A (pt) |
| LT (1) | LT3702371T (pt) |
| MA (1) | MA33208B1 (pt) |
| MX (2) | MX2011009961A (pt) |
| NZ (1) | NZ594339A (pt) |
| PE (1) | PE20120553A1 (pt) |
| RU (1) | RU2568066C2 (pt) |
| SG (1) | SG174904A1 (pt) |
| SI (2) | SI3702371T1 (pt) |
| TW (1) | TWI507205B (pt) |
| UA (1) | UA104459C2 (pt) |
| WO (1) | WO2010111367A1 (pt) |
| ZA (1) | ZA201105574B (pt) |
Families Citing this family (48)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| IL159177A0 (en) * | 2001-06-20 | 2004-06-01 | Prochon Biotech Ltd | Antibodies that block receptor protein tyrosine kinase activation, methods of screening for and uses thereof |
| JP5757534B2 (ja) | 2009-03-25 | 2015-07-29 | ジェネンテック, インコーポレイテッド | 抗fgfr3抗体およびこれを用いた方法 |
| EP2766000A2 (en) | 2011-10-15 | 2014-08-20 | F.Hoffmann-La Roche Ag | Scd1 antagonists for treating cancer |
| WO2013088191A1 (en) | 2011-12-12 | 2013-06-20 | Institut National De La Sante Et De La Recherche Medicale (Inserm) | Antagonist of the fibroblast growth factor receptor 3 (fgfr3) for use in the treatment or the prevention of skeletal disorders linked with abnormal activation of fgfr3 |
| US9714298B2 (en) * | 2012-04-09 | 2017-07-25 | Daiichi Sankyo Company, Limited | Anti-FGFR2 antibodies and methods of use thereof for treating cancer |
| EP2877854B1 (en) * | 2012-07-24 | 2022-09-07 | The Trustees of Columbia University in the City of New York | Fgfr-tacc fusion proteins and methods thereof |
| US20150203589A1 (en) | 2012-07-24 | 2015-07-23 | The Trustees Of Columbia University In The City Of New York | Fusion proteins and methods thereof |
| EP2877209A1 (en) * | 2012-07-27 | 2015-06-03 | Genentech, Inc. | Methods of treating fgfr3 related conditions |
| PL3489261T3 (pl) | 2012-08-24 | 2021-08-16 | The Regents Of The University Of California | Przeciwciała i szczepionki do stosowania w leczeniu nowotworów z ekspresją ROR1 i w hamowaniu przerzutu |
| CN104797710A (zh) | 2012-09-27 | 2015-07-22 | 中外制药株式会社 | Fgfr3融合基因和以其作为标靶的药物 |
| WO2014080004A1 (en) * | 2012-11-26 | 2014-05-30 | Santaris Pharma A/S | Compositions and methods for modulation of fgfr3 expression |
| US20140154702A1 (en) * | 2012-11-30 | 2014-06-05 | Endocyte, Inc. | Methods For Treating Cancer Using Combination Therapies |
| ES2705328T3 (es) * | 2012-12-21 | 2019-03-22 | Janssen Biotech Inc | Inmunoensayo sensible de múltiples etapas para receptores solubles de factor de crecimiento de fibroblastos |
| KR20160002681A (ko) | 2013-01-16 | 2016-01-08 | 인썸(인스티튜트 내셔날 드 라 싼테 에 드 라 리셰르셰메디칼르) | 골격 성장 지연 질환의 예방 또는 치료에 사용하기 위한 용해성 섬유아세포 성장 인자 수용체 3(fgr3) 폴리펩티드 |
| JP2016510751A (ja) | 2013-03-06 | 2016-04-11 | ジェネンテック, インコーポレイテッド | 抗がん剤耐性を治療及び予防する方法 |
| CN105792844B (zh) * | 2013-10-08 | 2021-03-02 | 第一三共株式会社 | 抗fgfr2抗体和另一药剂的组合 |
| WO2015164723A1 (en) | 2014-04-25 | 2015-10-29 | The Trustees Of The University Of Pennsylvania | Methods and compositions for treating metastatic breast cancer and other cancers in the brain |
| MA44179B1 (fr) | 2014-05-13 | 2020-10-28 | Univ Pennsylvania | Compositions comprenant un virus adeno-associe exprimant des constructions a double anticorps et leurs utilisations |
| WO2015191986A1 (en) | 2014-06-13 | 2015-12-17 | Genentech, Inc. | Methods of treating and preventing cancer drug resistance |
| US9573994B2 (en) | 2014-07-10 | 2017-02-21 | Bioarctic Neuroscience Ab | Aβ protofibril binding antibodies |
| EP3215533A2 (en) * | 2014-11-05 | 2017-09-13 | F. Hoffmann-La Roche AG | Anti-fgfr2/3 antibodies and methods using same |
| AU2015371312B2 (en) | 2014-12-24 | 2022-01-27 | Genentech, Inc. | Therapeutic, diagnostic and prognostic methods for cancer of the bladder |
| US20160243228A1 (en) * | 2015-02-19 | 2016-08-25 | Bioclin Therapeutics, Inc. | Methods, compositions, and kits for treatment of cancer |
| US20180237424A1 (en) | 2015-03-03 | 2018-08-23 | Inserm (Institut National De La Sante Et De La Recherche Medicale) | Fgfr3 antagonists |
| US11052133B2 (en) * | 2015-05-06 | 2021-07-06 | Protomer Technologies, Inc. | Glucose responsive insulins |
| CN108602890A (zh) | 2015-12-11 | 2018-09-28 | 瑞泽恩制药公司 | 用于减少或预防对egfr和/或erbb3阻滞剂具有抗性的肿瘤生长的方法 |
| AU2017239038A1 (en) | 2016-03-22 | 2018-10-04 | Bionomics Inc | Administration of an anti-LGR5 monoclonal antibody |
| CA3029003A1 (en) | 2016-06-27 | 2018-01-04 | The Regents Of The University Of California | Cancer treatment combinations |
| RU2751483C2 (ru) | 2016-07-07 | 2021-07-14 | Пфайзер Инк. | Полипептиды, являющиеся растворимыми рецепторами 3 фактора роста фибробластов (sfgfr3), и пути их применения |
| CA3048916A1 (en) * | 2017-02-06 | 2018-08-09 | Rainier Therapeutics, Inc. | Methods, compositions, and kits for treatment of cancer |
| US10722589B2 (en) | 2017-04-03 | 2020-07-28 | Covagen Ag | FGFR3 binding molecules |
| EP3634401A1 (en) * | 2017-06-07 | 2020-04-15 | Silverback Therapeutics, Inc. | Antibody construct conjugates |
| KR20210003099A (ko) * | 2018-03-19 | 2021-01-11 | 아베오미 코포레이션 | 세포예정사 리간드 1 (pd-l1)에 대한 고 친화도 중화 단일클론 항체 및 이의 용도 |
| US20200277387A1 (en) * | 2019-03-01 | 2020-09-03 | Rainier Therapeutics, Inc. | Methods and compositions for treating cancer |
| CN110272900B (zh) * | 2019-04-19 | 2024-03-26 | 中国人民解放军陆军军医大学 | 用于制备骨骼发育异常猪模型的sgRNA及其应用 |
| US20220267452A1 (en) | 2019-07-12 | 2022-08-25 | Chugai Seiyaku Kabushiki Kaisha | Anti-mutation type fgfr3 antibody and use therefor |
| CN114786730A (zh) * | 2019-11-05 | 2022-07-22 | 再生元制药公司 | N-端scFv多特异性结合分子 |
| AU2020387401A1 (en) * | 2019-11-18 | 2022-05-26 | Board Of Regents, The University Of Texas System | Anti-B7-H3 monoclonal antibody and methods of use thereof |
| AU2021327387A1 (en) * | 2020-08-21 | 2023-05-04 | Genzyme Corporation | Fgfr3 antibodies and methods of use |
| CA3202218A1 (en) | 2020-12-18 | 2022-06-23 | Century Therapeutics, Inc. | Chimeric antigen receptor systems with adaptable receptor specificity |
| CN114716548A (zh) * | 2021-01-05 | 2022-07-08 | (株)爱恩德生物 | 抗-fgfr3抗体及其用途 |
| WO2022221193A1 (en) | 2021-04-12 | 2022-10-20 | Affinia Therapeutics Inc. | Recombinant aav for treatment of neural disease |
| WO2023113806A1 (en) | 2021-12-16 | 2023-06-22 | Affinia Therapeutics, Inc. | Recombinant aav for treatment of neural disease |
| KR20240011714A (ko) | 2021-04-27 | 2024-01-26 | 제너레이션 바이오 컴퍼니 | 치료용 항체를 발현하는 비바이러스성 dna 벡터 및 이의 용도 |
| EP4329818A1 (en) | 2021-04-30 | 2024-03-06 | Celgene Corporation | Combination therapies using an anti-bcma antibody drug conjugate (adc) in combination with a gamma secretase inhibitor (gsi) |
| WO2023177655A1 (en) | 2022-03-14 | 2023-09-21 | Generation Bio Co. | Heterologous prime boost vaccine compositions and methods of use |
| WO2024002154A1 (en) * | 2022-06-28 | 2024-01-04 | Genequantum Healthcare (Suzhou) Co., Ltd. | Anti-fgfr3 antibody conjugate and medical use thereof |
| WO2024077095A1 (en) | 2022-10-05 | 2024-04-11 | Genentech, Inc. | Methods and compositions for classifying and treating bladder cancer |
Family Cites Families (165)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US510087A (en) | 1893-12-05 | Method of securing suspension-loops to the ends of hammock-bodies | ||
| US3773919A (en) | 1969-10-23 | 1973-11-20 | Du Pont | Polylactide-drug mixtures |
| US3896111A (en) | 1973-02-20 | 1975-07-22 | Research Corp | Ansa macrolides |
| US4151042A (en) | 1977-03-31 | 1979-04-24 | Takeda Chemical Industries, Ltd. | Method for producing maytansinol and its derivatives |
| US4137230A (en) | 1977-11-14 | 1979-01-30 | Takeda Chemical Industries, Ltd. | Method for the production of maytansinoids |
| USRE30985E (en) | 1978-01-01 | 1982-06-29 | Serum-free cell culture media | |
| FR2413974A1 (fr) | 1978-01-06 | 1979-08-03 | David Bernard | Sechoir pour feuilles imprimees par serigraphie |
| JPS54109494A (en) * | 1978-02-16 | 1979-08-28 | Teikoku Hormone Mfg Co Ltd | Method of measuring antigennantibody reaction |
| US4307016A (en) | 1978-03-24 | 1981-12-22 | Takeda Chemical Industries, Ltd. | Demethyl maytansinoids |
| US4265814A (en) | 1978-03-24 | 1981-05-05 | Takeda Chemical Industries | Matansinol 3-n-hexadecanoate |
| JPS5562090A (en) | 1978-10-27 | 1980-05-10 | Takeda Chem Ind Ltd | Novel maytansinoid compound and its preparation |
| US4256746A (en) | 1978-11-14 | 1981-03-17 | Takeda Chemical Industries | Dechloromaytansinoids, their pharmaceutical compositions and method of use |
| JPS55164687A (en) | 1979-06-11 | 1980-12-22 | Takeda Chem Ind Ltd | Novel maytansinoid compound and its preparation |
| JPS5566585A (en) * | 1978-11-14 | 1980-05-20 | Takeda Chem Ind Ltd | Novel maytansinoid compound and its preparation |
| JPS55102583A (en) | 1979-01-31 | 1980-08-05 | Takeda Chem Ind Ltd | 20-acyloxy-20-demethylmaytansinoid compound |
| JPS55162791A (en) * | 1979-06-05 | 1980-12-18 | Takeda Chem Ind Ltd | Antibiotic c-15003pnd and its preparation |
| JPS55164685A (en) | 1979-06-08 | 1980-12-22 | Takeda Chem Ind Ltd | Novel maytansinoid compound and its preparation |
| JPS55164686A (en) | 1979-06-11 | 1980-12-22 | Takeda Chem Ind Ltd | Novel maytansinoid compound and its preparation |
| US4309428A (en) | 1979-07-30 | 1982-01-05 | Takeda Chemical Industries, Ltd. | Maytansinoids |
| JPS5645483A (en) | 1979-09-19 | 1981-04-25 | Takeda Chem Ind Ltd | C-15003phm and its preparation |
| JPS5645485A (en) | 1979-09-21 | 1981-04-25 | Takeda Chem Ind Ltd | Production of c-15003pnd |
| EP0028683A1 (en) | 1979-09-21 | 1981-05-20 | Takeda Chemical Industries, Ltd. | Antibiotic C-15003 PHO and production thereof |
| WO1982001188A1 (en) | 1980-10-08 | 1982-04-15 | Takeda Chemical Industries Ltd | 4,5-deoxymaytansinoide compounds and process for preparing same |
| US4450254A (en) | 1980-11-03 | 1984-05-22 | Standard Oil Company | Impact improvement of high nitrile resins |
| US4419446A (en) | 1980-12-31 | 1983-12-06 | The United States Of America As Represented By The Department Of Health And Human Services | Recombinant DNA process utilizing a papilloma virus DNA as a vector |
| US4315929A (en) | 1981-01-27 | 1982-02-16 | The United States Of America As Represented By The Secretary Of Agriculture | Method of controlling the European corn borer with trewiasine |
| US4313946A (en) | 1981-01-27 | 1982-02-02 | The United States Of America As Represented By The Secretary Of Agriculture | Chemotherapeutically active maytansinoids from Trewia nudiflora |
| JPS57192389A (en) | 1981-05-20 | 1982-11-26 | Takeda Chem Ind Ltd | Novel maytansinoid |
| US4601978A (en) * | 1982-11-24 | 1986-07-22 | The Regents Of The University Of California | Mammalian metallothionein promoter system |
| US4560655A (en) | 1982-12-16 | 1985-12-24 | Immunex Corporation | Serum-free cell culture medium and process for making same |
| US4657866A (en) | 1982-12-21 | 1987-04-14 | Sudhir Kumar | Serum-free, synthetic, completely chemically defined tissue culture media |
| US4816567A (en) | 1983-04-08 | 1989-03-28 | Genentech, Inc. | Recombinant immunoglobin preparations |
| US4675187A (en) | 1983-05-16 | 1987-06-23 | Bristol-Myers Company | BBM-1675, a new antibiotic complex |
| US4767704A (en) * | 1983-10-07 | 1988-08-30 | Columbia University In The City Of New York | Protein-free culture medium |
| US4683203A (en) | 1984-04-14 | 1987-07-28 | Redco N.V. | Immobilized enzymes, processes for preparing same, and use thereof |
| US4965199A (en) * | 1984-04-20 | 1990-10-23 | Genentech, Inc. | Preparation of functional human factor VIII in mammalian cells using methotrexate based selection |
| GB8426467D0 (en) | 1984-10-19 | 1984-11-28 | Technology Licence Co Ltd | Monoclonal antibodies |
| US4970198A (en) | 1985-10-17 | 1990-11-13 | American Cyanamid Company | Antitumor antibiotics (LL-E33288 complex) |
| US4683195A (en) | 1986-01-30 | 1987-07-28 | Cetus Corporation | Process for amplifying, detecting, and/or-cloning nucleic acid sequences |
| GB8516415D0 (en) | 1985-06-28 | 1985-07-31 | Celltech Ltd | Culture of animal cells |
| US4676980A (en) | 1985-09-23 | 1987-06-30 | The United States Of America As Represented By The Secretary Of The Department Of Health And Human Services | Target specific cross-linked heteroantibodies |
| US4927762A (en) | 1986-04-01 | 1990-05-22 | Cell Enterprises, Inc. | Cell culture medium with antioxidant |
| IL85035A0 (en) | 1987-01-08 | 1988-06-30 | Int Genetic Eng | Polynucleotide molecule,a chimeric antibody with specificity for human b cell surface antigen,a process for the preparation and methods utilizing the same |
| US5079233A (en) | 1987-01-30 | 1992-01-07 | American Cyanamid Company | N-acyl derivatives of the LL-E33288 antitumor antibiotics, composition and methods for using the same |
| JP3101690B2 (ja) * | 1987-03-18 | 2000-10-23 | エス・ビィ・2・インコーポレイテッド | 変性抗体の、または変性抗体に関する改良 |
| US4975278A (en) | 1988-02-26 | 1990-12-04 | Bristol-Myers Company | Antibody-enzyme conjugates in combination with prodrugs for the delivery of cytotoxic agents to tumor cells |
| US5053394A (en) | 1988-09-21 | 1991-10-01 | American Cyanamid Company | Targeted forms of methyltrithio antitumor agents |
| US5770701A (en) * | 1987-10-30 | 1998-06-23 | American Cyanamid Company | Process for preparing targeted forms of methyltrithio antitumor agents |
| US5606040A (en) | 1987-10-30 | 1997-02-25 | American Cyanamid Company | Antitumor and antibacterial substituted disulfide derivatives prepared from compounds possessing a methyl-trithio group |
| IE61148B1 (en) | 1988-03-10 | 1994-10-05 | Ici Plc | Method of detecting nucleotide sequences |
| US20030232010A2 (en) * | 1988-03-29 | 2003-12-18 | Immunomedics, Inc. | Improved cytotoxic therapy |
| EP0435911B1 (en) | 1988-09-23 | 1996-03-13 | Cetus Oncology Corporation | Cell culture medium for enhanced cell growth, culture longevity and product expression |
| US5530101A (en) | 1988-12-28 | 1996-06-25 | Protein Design Labs, Inc. | Humanized immunoglobulins |
| DE3920358A1 (de) | 1989-06-22 | 1991-01-17 | Behringwerke Ag | Bispezifische und oligospezifische, mono- und oligovalente antikoerperkonstrukte, ihre herstellung und verwendung |
| ES2096590T3 (es) | 1989-06-29 | 1997-03-16 | Medarex Inc | Reactivos biespecificos para la terapia del sida. |
| HU215581B (hu) * | 1989-07-06 | 1999-01-28 | Regents Of The University Of California | Eljárás fibroblaszt növekedési faktor receptorok előállítására és terápiás alkalmazására |
| CA2026147C (en) | 1989-10-25 | 2006-02-07 | Ravi J. Chari | Cytotoxic agents comprising maytansinoids and their therapeutic use |
| US5208020A (en) | 1989-10-25 | 1993-05-04 | Immunogen Inc. | Cytotoxic agents comprising maytansinoids and their therapeutic use |
| WO1992000373A1 (en) | 1990-06-29 | 1992-01-09 | Biosource Genetics Corporation | Melanin production by transformed microorganisms |
| US5122469A (en) | 1990-10-03 | 1992-06-16 | Genentech, Inc. | Method for culturing Chinese hamster ovary cells to improve production of recombinant proteins |
| US5264365A (en) | 1990-11-09 | 1993-11-23 | Board Of Regents, The University Of Texas System | Protease-deficient bacterial strains for production of proteolytically sensitive polypeptides |
| US5508192A (en) * | 1990-11-09 | 1996-04-16 | Board Of Regents, The University Of Texas System | Bacterial host strains for producing proteolytically sensitive polypeptides |
| DK0564531T3 (da) | 1990-12-03 | 1998-09-28 | Genentech Inc | Berigelsesfremgangsmåde for variantproteiner med ændrede bindingsegenskaber |
| US5571894A (en) | 1991-02-05 | 1996-11-05 | Ciba-Geigy Corporation | Recombinant antibodies specific for a growth factor receptor |
| MX9202773A (es) | 1991-06-10 | 1993-02-01 | Keystone Int | Metodo y aparato para monitorear el funcionamiento del sistema de control de recirculacion. |
| LU91067I2 (fr) | 1991-06-14 | 2004-04-02 | Genentech Inc | Trastuzumab et ses variantes et dérivés immuno chimiques y compris les immotoxines |
| CA2116774C (en) | 1991-09-19 | 2003-11-11 | Paul J. Carter | Expression in e. coli antibody fragments having at least a cysteine present as a free thiol. use for the production of bifunctional f(ab') 2 antibodies |
| WO1993006213A1 (en) | 1991-09-23 | 1993-04-01 | Medical Research Council | Production of chimeric antibodies - a combinatorial approach |
| US5362852A (en) | 1991-09-27 | 1994-11-08 | Pfizer Inc. | Modified peptide derivatives conjugated at 2-hydroxyethylamine moieties |
| US5587458A (en) | 1991-10-07 | 1996-12-24 | Aronex Pharmaceuticals, Inc. | Anti-erbB-2 antibodies, combinations thereof, and therapeutic and diagnostic uses thereof |
| WO1993008829A1 (en) | 1991-11-04 | 1993-05-13 | The Regents Of The University Of California | Compositions that mediate killing of hiv-infected cells |
| DE69233528T2 (de) | 1991-11-25 | 2006-03-16 | Enzon, Inc. | Verfahren zur Herstellung von multivalenten antigenbindenden Proteinen |
| IL100219A0 (en) | 1991-12-02 | 1992-09-06 | Yeda Res & Dev | Variable region within fibroblast growth factor receptors that confers ligand specificity |
| US5667988A (en) | 1992-01-27 | 1997-09-16 | The Scripps Research Institute | Methods for producing antibody libraries using universal or randomized immunoglobulin light chains |
| CA2372813A1 (en) | 1992-02-06 | 1993-08-19 | L.L. Houston | Biosynthetic binding protein for cancer marker |
| ZA932522B (en) | 1992-04-10 | 1993-12-20 | Res Dev Foundation | Immunotoxins directed against c-erbB-2(HER/neu) related surface antigens |
| DE69308573T2 (de) | 1992-08-17 | 1997-08-07 | Genentech Inc | Bispezifische immunoadhesine |
| JP3095175B2 (ja) | 1992-11-13 | 2000-10-03 | アイデック ファーマシューティカルズ コーポレイション | B細胞リンパ腫の治療のためのヒトbリンパ球限定分化抗原に対するキメラ抗体と放射能標識抗体の療法利用 |
| US5635483A (en) | 1992-12-03 | 1997-06-03 | Arizona Board Of Regents Acting On Behalf Of Arizona State University | Tumor inhibiting tetrapeptide bearing modified phenethyl amides |
| US5780588A (en) | 1993-01-26 | 1998-07-14 | Arizona Board Of Regents | Elucidation and synthesis of selected pentapeptides |
| AU6446194A (en) | 1993-03-17 | 1994-10-11 | Whittier Institute For Diabetes And Endocrinology, The | Monoclonal antibodies specific for fibroblast growth factor receptors, immunotoxins, and use thereof |
| WO1994029351A2 (en) | 1993-06-16 | 1994-12-22 | Celltech Limited | Antibodies |
| US5773001A (en) | 1994-06-03 | 1998-06-30 | American Cyanamid Company | Conjugates of methyltrithio antitumor agents and intermediates for their synthesis |
| US5910486A (en) | 1994-09-06 | 1999-06-08 | Uab Research Foundation | Methods for modulating protein function in cells using, intracellular antibody homologues |
| US5639635A (en) | 1994-11-03 | 1997-06-17 | Genentech, Inc. | Process for bacterial production of polypeptides |
| US5663149A (en) * | 1994-12-13 | 1997-09-02 | Arizona Board Of Regents Acting On Behalf Of Arizona State University | Human cancer inhibitory pentapeptide heterocyclic and halophenyl amides |
| US5641870A (en) | 1995-04-20 | 1997-06-24 | Genentech, Inc. | Low pH hydrophobic interaction chromatography for antibody purification |
| US5869046A (en) | 1995-04-14 | 1999-02-09 | Genentech, Inc. | Altered polypeptides with increased half-life |
| US5739277A (en) | 1995-04-14 | 1998-04-14 | Genentech Inc. | Altered polypeptides with increased half-life |
| US5712374A (en) * | 1995-06-07 | 1998-01-27 | American Cyanamid Company | Method for the preparation of substantiallly monomeric calicheamicin derivative/carrier conjugates |
| US5714586A (en) | 1995-06-07 | 1998-02-03 | American Cyanamid Company | Methods for the preparation of monomeric calicheamicin derivative/carrier conjugates |
| DE69619526T2 (de) | 1995-06-12 | 2002-10-31 | Yeda Res & Dev | Fgf9 als spezifischer ligand für fgfr3 |
| GB9603256D0 (en) | 1996-02-16 | 1996-04-17 | Wellcome Found | Antibodies |
| US6027888A (en) | 1996-04-05 | 2000-02-22 | Board Of Regents, The University Of Texas System | Methods for producing soluble, biologically-active disulfide-bond containing eukaryotic proteins in bacterial cells |
| US5869245A (en) * | 1996-06-05 | 1999-02-09 | Fox Chase Cancer Center | Mismatch endonuclease and its use in identifying mutations in targeted polynucleotide strands |
| US5994071A (en) | 1997-04-04 | 1999-11-30 | Albany Medical College | Assessment of prostate cancer |
| US6083715A (en) | 1997-06-09 | 2000-07-04 | Board Of Regents, The University Of Texas System | Methods for producing heterologous disulfide bond-containing polypeptides in bacterial cells |
| US6306591B1 (en) * | 1997-06-18 | 2001-10-23 | Utah State University | Screening for the molecular defect causing spider lamb syndrome in sheep |
| WO1998058964A1 (en) | 1997-06-24 | 1998-12-30 | Genentech, Inc. | Methods and compositions for galactosylated glycoproteins |
| WO1999022764A1 (en) | 1997-10-31 | 1999-05-14 | Genentech, Inc. | Methods and compositions comprising glycoprotein glycoforms |
| US6194551B1 (en) * | 1998-04-02 | 2001-02-27 | Genentech, Inc. | Polypeptide variants |
| CA2323757C (en) | 1998-04-02 | 2011-08-02 | Genentech, Inc. | Antibody variants and fragments thereof |
| DK1071700T3 (da) | 1998-04-20 | 2010-06-07 | Glycart Biotechnology Ag | Glykosylerings-modifikation af antistoffer til forbedring af antistofafhængig cellulær cytotoksicitet |
| US6849255B2 (en) | 1998-08-18 | 2005-02-01 | Yissum Research Development Company Of The Hebrew University Of Jerusalem | Methods and compositions for enhancing cartilage repair |
| EP1141024B1 (en) | 1999-01-15 | 2018-08-08 | Genentech, Inc. | POLYPEPTIDE COMPRISING A VARIANT HUMAN IgG1 Fc REGION |
| US6737056B1 (en) | 1999-01-15 | 2004-05-18 | Genentech, Inc. | Polypeptide variants with altered effector function |
| IL128380A0 (en) * | 1999-02-04 | 2000-01-31 | Yeda Res & Dev | A method of screening for agonists and antagonists of FGFR |
| DK2270150T4 (da) | 1999-04-09 | 2019-08-26 | Kyowa Hakko Kirin Co Ltd | Fremgangsmåde til at kontrollere aktiviteten af immunologisk funktionelt molekyle. |
| US7135311B1 (en) | 1999-05-05 | 2006-11-14 | Institut Curie | Means for detecting and treating pathologies linked to FGFR3 |
| CA2388245C (en) | 1999-10-19 | 2012-01-10 | Tatsuya Ogawa | The use of serum-free adapted rat cells for producing heterologous polypeptides |
| EP2180054A1 (en) | 1999-12-24 | 2010-04-28 | Genentech, Inc. | Methods and compositions for prolonging elimination half-times of bioactive compounds |
| US6946292B2 (en) | 2000-10-06 | 2005-09-20 | Kyowa Hakko Kogyo Co., Ltd. | Cells producing antibody compositions with increased antibody dependent cytotoxic activity |
| US7064191B2 (en) * | 2000-10-06 | 2006-06-20 | Kyowa Hakko Kogyo Co., Ltd. | Process for purifying antibody |
| IL139380A0 (en) | 2000-10-31 | 2001-11-25 | Prochon Biotech Ltd | Active variants of fibroblast growth factor |
| PT1355919E (pt) | 2000-12-12 | 2011-03-02 | Medimmune Llc | Moléculas com semivida longa, composições que as contêm e suas utilizações |
| CA2432257A1 (en) | 2000-12-18 | 2002-06-27 | Gene Logic, Inc. | Treatment of bone disorders by modulation of fgfr3 |
| US20040116330A1 (en) | 2001-04-27 | 2004-06-17 | Kenichiro Naito | Preventive/therapeutic method for cancer |
| US6884869B2 (en) | 2001-04-30 | 2005-04-26 | Seattle Genetics, Inc. | Pentapeptide compounds and uses related thereto |
| IL159177A0 (en) | 2001-06-20 | 2004-06-01 | Prochon Biotech Ltd | Antibodies that block receptor protein tyrosine kinase activation, methods of screening for and uses thereof |
| KR20100018071A (ko) | 2001-08-03 | 2010-02-16 | 글리카트 바이오테크놀로지 아게 | 항체 의존적 세포 독성이 증가된 항체 글리코실화 변이체 |
| KR100988949B1 (ko) | 2001-10-25 | 2010-10-20 | 제넨테크, 인크. | 당단백질 조성물 |
| US20040093621A1 (en) | 2001-12-25 | 2004-05-13 | Kyowa Hakko Kogyo Co., Ltd | Antibody composition which specifically binds to CD20 |
| JPWO2003084569A1 (ja) * | 2002-04-09 | 2005-08-11 | 協和醗酵工業株式会社 | 抗体組成物含有医薬 |
| US7749753B2 (en) | 2002-04-09 | 2010-07-06 | Kyowa Hakko Kirin Co., Ltd | Cells in which activity of the protein involved in transportation of GDP-fucose is reduced or lost |
| CA2481657A1 (en) | 2002-04-09 | 2003-10-16 | Kyowa Hakko Kogyo Co., Ltd. | Cells of which genome is modified |
| US20050031613A1 (en) | 2002-04-09 | 2005-02-10 | Kazuyasu Nakamura | Therapeutic agent for patients having human FcgammaRIIIa |
| AU2003236018A1 (en) | 2002-04-09 | 2003-10-20 | Kyowa Hakko Kirin Co., Ltd. | METHOD OF ENHANCING ACTIVITY OF ANTIBODY COMPOSITION OF BINDING TO FcGamma RECEPTOR IIIa |
| JPWO2003085118A1 (ja) | 2002-04-09 | 2005-08-11 | 協和醗酵工業株式会社 | 抗体組成物の製造方法 |
| EP1391213A1 (en) | 2002-08-21 | 2004-02-25 | Boehringer Ingelheim International GmbH | Compositions and methods for treating cancer using maytansinoid CD44 antibody immunoconjugates and chemotherapeutic agents |
| US7361740B2 (en) * | 2002-10-15 | 2008-04-22 | Pdl Biopharma, Inc. | Alteration of FcRn binding affinities or serum half-lives of antibodies by mutagenesis |
| ES2347241T3 (es) | 2002-12-16 | 2010-10-27 | Genentech, Inc. | Variantes de inmunoglobulina y sus utilizaciones. |
| DK1611252T3 (da) * | 2003-03-26 | 2009-11-23 | Progenika Biopharma Sa | In vitro-fremgangsmåde til påvisning af overgangscelle-karcinom i blæren |
| US8088387B2 (en) * | 2003-10-10 | 2012-01-03 | Immunogen Inc. | Method of targeting specific cell populations using cell-binding agent maytansinoid conjugates linked via a non-cleavable linker, said conjugates, and methods of making said conjugates |
| IL156495A0 (en) | 2003-06-17 | 2004-01-04 | Prochon Biotech Ltd | Use of fgfr3 antagonists for treating t cell mediated diseases |
| EP1688439A4 (en) | 2003-10-08 | 2007-12-19 | Kyowa Hakko Kogyo Kk | HYBRID PROTEIN COMPOSITION |
| AU2004280065A1 (en) | 2003-10-09 | 2005-04-21 | Kyowa Hakko Kirin Co., Ltd. | Process for producing antibody composition by using RNA inhibiting the function of alpha1,6-fucosyltransferase |
| US9296820B2 (en) | 2003-11-05 | 2016-03-29 | Roche Glycart Ag | Polynucleotides encoding anti-CD20 antigen binding molecules with increased Fc receptor binding affinity and effector function |
| ES2697327T3 (es) | 2003-11-06 | 2019-01-23 | Seattle Genetics Inc | Compuesto intermedio para la preparación de conjugados que comprenden derivados de auristatina y un enlazador |
| WO2005053742A1 (ja) | 2003-12-04 | 2005-06-16 | Kyowa Hakko Kogyo Co., Ltd. | 抗体組成物を含有する医薬 |
| CA2550245A1 (en) * | 2003-12-19 | 2005-07-21 | Five Prime Therapeutics, Inc. | Fibroblast growth factor receptors 1, 2, 3, and 4 as targets for therapeutic intervention |
| RS51326B (sr) | 2004-08-05 | 2010-12-31 | Genentech Inc. | Humanizovani anti-cmet antagonisti |
| AU2005302083A1 (en) | 2004-11-04 | 2006-05-11 | Fibron Limited | Treatment of B-cell malignancies |
| TW200902555A (en) * | 2005-01-03 | 2009-01-16 | Hoffmann La Roche | Antibodies against IL-13 receptor alpha 1 and uses thereof |
| JP5196654B2 (ja) | 2005-06-20 | 2013-05-15 | ジェネンテック, インコーポレイテッド | 腫瘍の診断と治療のための組成物と方法 |
| JP5102771B2 (ja) | 2005-10-28 | 2012-12-19 | アイアールエム・リミテッド・ライアビリティ・カンパニー | タンパク質キナーゼとしての化合物および組成物 |
| CA2638785C (en) | 2006-01-05 | 2017-02-21 | Genentech, Inc. | Anti-ephb4 antibodies and methods using same |
| US8101721B2 (en) * | 2006-06-15 | 2012-01-24 | Fibron Ltd. | Antibodies blocking fibroblast growth factor receptor activation and methods of use thereof |
| US8153764B2 (en) | 2007-03-15 | 2012-04-10 | Reverse Proteomics Research Institute Co., Ltd. | Biomarker specific to brain/nerve or specific to neuronal differentiation |
| US8933202B2 (en) | 2007-11-12 | 2015-01-13 | U3 Pharma Gmbh | AXL antibodies |
| UY31800A (es) | 2008-05-05 | 2009-11-10 | Smithkline Beckman Corp | Metodo de tratamiento de cancer usando un inhibidor de cmet y axl y un inhibidor de erbb |
| US8187601B2 (en) * | 2008-07-01 | 2012-05-29 | Aveo Pharmaceuticals, Inc. | Fibroblast growth factor receptor 3 (FGFR3) binding proteins |
| WO2010098788A2 (en) | 2008-08-25 | 2010-09-02 | Amplimmune, Inc. | Pd-i antagonists and methods for treating infectious disease |
| TWI381848B (zh) * | 2008-10-20 | 2013-01-11 | Imclone Llc | 抗纖維母細胞生長因子受體-3 (fgfr-3) 抗體及包含其之醫藥組合物 |
| JP5757534B2 (ja) | 2009-03-25 | 2015-07-29 | ジェネンテック, インコーポレイテッド | 抗fgfr3抗体およびこれを用いた方法 |
| US20130017199A1 (en) | 2009-11-24 | 2013-01-17 | AMPLIMMUNE ,Inc. a corporation | Simultaneous inhibition of pd-l1/pd-l2 |
| WO2012088266A2 (en) | 2010-12-22 | 2012-06-28 | Incyte Corporation | Substituted imidazopyridazines and benzimidazoles as inhibitors of fgfr3 |
| UY34887A (es) | 2012-07-02 | 2013-12-31 | Bristol Myers Squibb Company Una Corporacion Del Estado De Delaware | Optimización de anticuerpos que se fijan al gen de activación de linfocitos 3 (lag-3) y sus usos |
| EP2877209A1 (en) | 2012-07-27 | 2015-06-03 | Genentech, Inc. | Methods of treating fgfr3 related conditions |
| EP2743657B1 (en) | 2012-12-13 | 2016-03-02 | Thomson Licensing | Method for controlling an image display device to allow an observer to perceive colours as another observer |
| RU2019129525A (ru) | 2013-07-16 | 2019-11-05 | Дженентек, Инк. | Способы лечения рака с использованием антагонистов, связывающих с осью pd-1, и ингибиторов tigit |
| TWI541022B (zh) | 2013-12-18 | 2016-07-11 | 應克隆公司 | 針對纖維母細胞生長因子受體-3(fgfr3)之化合物及治療方法 |
| US20160243228A1 (en) | 2015-02-19 | 2016-08-25 | Bioclin Therapeutics, Inc. | Methods, compositions, and kits for treatment of cancer |
| EP3398007A1 (en) | 2016-02-04 | 2018-11-07 | DigiLens, Inc. | Holographic waveguide optical tracker |
| CA3048916A1 (en) | 2017-02-06 | 2018-08-09 | Rainier Therapeutics, Inc. | Methods, compositions, and kits for treatment of cancer |
| US20200277387A1 (en) | 2019-03-01 | 2020-09-03 | Rainier Therapeutics, Inc. | Methods and compositions for treating cancer |
-
2010
- 2010-03-24 JP JP2012502202A patent/JP5757534B2/ja active Active
- 2010-03-24 MA MA34269A patent/MA33208B1/fr unknown
- 2010-03-24 CA CA2754163A patent/CA2754163C/en active Active
- 2010-03-24 AU AU2010229994A patent/AU2010229994B2/en not_active Ceased
- 2010-03-24 SG SG2011068897A patent/SG174904A1/en unknown
- 2010-03-24 ES ES10722819.9T patent/ES2549769T3/es active Active
- 2010-03-24 NZ NZ594339A patent/NZ594339A/xx unknown
- 2010-03-24 HU HUE10722819A patent/HUE025726T2/en unknown
- 2010-03-24 CN CN201080013816.8A patent/CN102378767B/zh active Active
- 2010-03-24 MX MX2011009961A patent/MX2011009961A/es active IP Right Grant
- 2010-03-24 EP EP10722819.9A patent/EP2411414B1/en active Active
- 2010-03-24 EP EP20130183384 patent/EP2679600A1/en not_active Ceased
- 2010-03-24 DK DK19175988.5T patent/DK3702371T3/da active
- 2010-03-24 HR HRP20221471TT patent/HRP20221471T1/hr unknown
- 2010-03-24 LT LTEP19175988.5T patent/LT3702371T/lt unknown
- 2010-03-24 UA UAA201112490A patent/UA104459C2/uk unknown
- 2010-03-24 HU HUE19175988A patent/HUE061117T2/hu unknown
- 2010-03-24 EP EP19175988.5A patent/EP3702371B1/en active Active
- 2010-03-24 PE PE2011001694A patent/PE20120553A1/es not_active Application Discontinuation
- 2010-03-24 WO PCT/US2010/028470 patent/WO2010111367A1/en active Application Filing
- 2010-03-24 DK DK10722819.9T patent/DK2411414T3/en active
- 2010-03-24 BR BRPI1006448-6A patent/BRPI1006448B1/pt active IP Right Grant
- 2010-03-24 KR KR1020117025047A patent/KR101822663B1/ko active IP Right Grant
- 2010-03-24 SI SI201032125T patent/SI3702371T1/sl unknown
- 2010-03-24 US US12/661,852 patent/US20100291114A1/en not_active Abandoned
- 2010-03-24 US US12/731,100 patent/US8410250B2/en active Active
- 2010-03-24 CN CN201510080510.9A patent/CN104788564A/zh active Pending
- 2010-03-24 TW TW099108751A patent/TWI507205B/zh active
- 2010-03-24 ES ES19175988T patent/ES2932874T3/es active Active
- 2010-03-24 KR KR1020167032334A patent/KR101830024B1/ko active IP Right Grant
- 2010-03-24 RU RU2011142905/10A patent/RU2568066C2/ru active
- 2010-03-24 SI SI201031035T patent/SI2411414T1/sl unknown
- 2010-03-24 MX MX2014012836A patent/MX345909B/es unknown
- 2010-03-25 AR ARP100100947A patent/AR075939A1/es active IP Right Grant
-
2011
- 2011-07-28 ZA ZA2011/05574A patent/ZA201105574B/en unknown
- 2011-08-03 IL IL214433A patent/IL214433A/en active IP Right Grant
- 2011-08-28 EC EC2011011417A patent/ECSP11011417A/es unknown
- 2011-09-01 CO CO11112595A patent/CO6450665A2/es active IP Right Grant
-
2012
- 2012-08-10 US US13/572,557 patent/US20120321606A1/en not_active Abandoned
- 2012-08-24 HK HK12108307.7A patent/HK1167666A1/xx unknown
- 2012-10-16 US US13/653,293 patent/US8710189B2/en active Active
-
2013
- 2013-02-07 US US13/762,252 patent/US9161977B2/en active Active
-
2014
- 2014-03-04 US US14/197,072 patent/US20140187754A1/en not_active Abandoned
-
2015
- 2015-09-14 HR HRP20150965TT patent/HRP20150965T1/hr unknown
- 2015-10-19 US US14/887,042 patent/US9499623B2/en active Active
-
2016
- 2016-09-15 US US15/266,105 patent/US10000571B2/en active Active
- 2016-11-18 US US15/356,483 patent/US10287356B2/en active Active
-
2017
- 2017-02-15 IL IL250624A patent/IL250624B/en active IP Right Grant
-
2019
- 2019-04-10 US US16/380,865 patent/US11401333B2/en active Active
-
2020
- 2020-08-12 AR ARP200102285A patent/AR119754A2/es unknown
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| US11401333B2 (en) | Anti-FGFR3 antibodies and methods using same | |
| US10208120B2 (en) | Anti-FGFR2/3 antibodies and methods using same | |
| JP2012521759A5 (pt) | ||
| JP2017538442A5 (pt) | ||
| AU2013205318B2 (en) | Anti-FGFR3 antibodies and methods using same |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| B06F | Objections, documents and/or translations needed after an examination request according [chapter 6.6 patent gazette] | ||
| B07D | Technical examination (opinion) related to article 229 of industrial property law [chapter 7.4 patent gazette] |
Free format text: DE ACORDO COM O ARTIGO 229-C DA LEI NO 10196/2001, QUE MODIFICOU A LEI NO 9279/96, A CONCESSAO DA PATENTE ESTA CONDICIONADA A ANUENCIA PREVIA DA ANVISA. CONSIDERANDO A APROVACAO DOS TERMOS DO PARECER NO 337/PGF/EA/2010, BEM COMO A PORTARIA INTERMINISTERIAL NO 1065 DE 24/05/2012, ENCAMINHA-SE O PRESENTE PEDIDO PARA AS PROVIDENCIAS CABIVEIS. |
|
| B07E | Notification of approval relating to section 229 industrial property law [chapter 7.5 patent gazette] | ||
| B06U | Preliminary requirement: requests with searches performed by other patent offices: procedure suspended [chapter 6.21 patent gazette] | ||
| B09A | Decision: intention to grant [chapter 9.1 patent gazette] | ||
| B16A | Patent or certificate of addition of invention granted [chapter 16.1 patent gazette] |
Free format text: PRAZO DE VALIDADE: 20 (VINTE) ANOS CONTADOS A PARTIR DE 24/03/2010, OBSERVADAS AS CONDICOES LEGAIS. PATENTE CONCEDIDA CONFORME ADI 5.529/DF, QUE DETERMINA A ALTERACAO DO PRAZO DE CONCESSAO. |