WO2010104092A1 - 植物のバイオマス量及び/又は種子量を増産させる遺伝子及びその利用方法 - Google Patents
植物のバイオマス量及び/又は種子量を増産させる遺伝子及びその利用方法 Download PDFInfo
- Publication number
- WO2010104092A1 WO2010104092A1 PCT/JP2010/053939 JP2010053939W WO2010104092A1 WO 2010104092 A1 WO2010104092 A1 WO 2010104092A1 JP 2010053939 W JP2010053939 W JP 2010053939W WO 2010104092 A1 WO2010104092 A1 WO 2010104092A1
- Authority
- WO
- WIPO (PCT)
- Prior art keywords
- gene
- plant
- amino acid
- seq
- acid sequence
- Prior art date
Links
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/82—Vectors or expression systems specially adapted for eukaryotic hosts for plant cells, e.g. plant artificial chromosomes (PACs)
- C12N15/8241—Phenotypically and genetically modified plants via recombinant DNA technology
- C12N15/8261—Phenotypically and genetically modified plants via recombinant DNA technology with agronomic (input) traits, e.g. crop yield
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/14—Hydrolases (3)
- C12N9/16—Hydrolases (3) acting on ester bonds (3.1)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/90—Isomerases (5.)
-
- Y—GENERAL TAGGING OF NEW TECHNOLOGICAL DEVELOPMENTS; GENERAL TAGGING OF CROSS-SECTIONAL TECHNOLOGIES SPANNING OVER SEVERAL SECTIONS OF THE IPC; TECHNICAL SUBJECTS COVERED BY FORMER USPC CROSS-REFERENCE ART COLLECTIONS [XRACs] AND DIGESTS
- Y02—TECHNOLOGIES OR APPLICATIONS FOR MITIGATION OR ADAPTATION AGAINST CLIMATE CHANGE
- Y02A—TECHNOLOGIES FOR ADAPTATION TO CLIMATE CHANGE
- Y02A40/00—Adaptation technologies in agriculture, forestry, livestock or agroalimentary production
- Y02A40/10—Adaptation technologies in agriculture, forestry, livestock or agroalimentary production in agriculture
- Y02A40/146—Genetically Modified [GMO] plants, e.g. transgenic plants
Definitions
- the present invention includes a plant into which a predetermined gene has been introduced or a modified expression control region of the endogenous gene, and a biomass amount by introducing the predetermined gene or modifying the endogenous gene expression control region
- the present invention also relates to a method for increasing the amount of seeds and / or a method for producing a plant having increased amounts of biomass and / or seeds.
- Biomass generally refers to the total amount of organisms that inhabit or exist per certain area, and in particular when plants are targeted, it means the dry weight per unit area.
- the unit of biomass is quantified by mass or energy amount.
- biomass and “biomass” are synonymous with the expression “biomass”.
- Standing crop is sometimes used. Since plant biomass is generated by fixing carbon dioxide in the atmosphere using solar energy, it can be captured as so-called carbon neutral energy. Therefore, increasing plant biomass has the effects of global environmental conservation, prevention of global warming, and reduction of greenhouse gas emissions. Therefore, the technology for increasing plant biomass production is highly industrially important.
- plants are cultivated for the purpose of part of the tissue itself (seed, root, leaf stem, etc.) or for the purpose of producing various substances such as fats and oils.
- fats and oils produced by plants soybean oil, sesame oil, olive oil, coconut oil, rice oil, cottonseed oil, sunflower oil, corn oil, bean flower oil, palm oil, rapeseed oil, etc. have been known since ancient times. Widely used in industrial applications.
- oils and fats produced by plants are also used as raw materials for biodiesel fuel and bioplastics, and their applicability is expanding as petroleum alternative energy.
- Patent Documents 1 to 7 As a technique for increasing plant biomass production, for example, various gene transfer techniques disclosed in Patent Documents 1 to 7 have been known. However, none of the technologies is sufficient for increasing biomass production.
- Patent Document 8 discloses a transformed plant having improved growth and disease resistance by overexpressing a glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene (FBA1 gene). .
- FBA1 gene glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene
- an object of the present invention is to search for a gene having a novel function for greatly improving the amount of plant biomass and to provide a technique capable of greatly increasing the amount of plant biomass.
- the plant according to the present invention comprises a gene encoding protein phosphatase 2C having three common sequences consisting of the amino acid sequences shown in SEQ ID NOs: 1 to 3 in this order and glutathione-binding plastid type fructose 1, A gene encoding 6-bisphosphate aldolase is introduced, or the expression control region of the gene is modified.
- the method for increasing the production of biomass comprises a gene encoding protein phosphatase 2C having three common sequences consisting of amino acid sequences shown in SEQ ID NOs: 1 to 3 in this order from the N-terminal side, and a glutathione-binding plastid.
- This is a method of introducing a gene encoding type fructose 1,6-bisphosphate aldolase or modifying the expression control region of the gene concerned.
- the method for producing a plant according to the present invention comprises a gene encoding protein phosphatase 2C having three common sequences consisting of amino acid sequences shown in SEQ ID NOs: 1 to 3 in this order from the N-terminal side, and a glutathione-binding plastid type.
- the gene encoding the protein phosphatase 2C is At1g03590-AtPP2C6-6, At1g16220, At1g79630, At5g01700, At3g02750, At5g36250, At5g26010, At4g32950, At3g16800, At3g05640, At5g27930-At2g2270 At least one kind of gene selected from or a gene functionally equivalent to the gene.
- the gene encoding the protein phosphatase 2C encodes one of the following proteins (a) to (c).
- (b) a protein phosphatase 2C activity comprising an amino acid sequence in which one or more amino acids are deleted, substituted, added or inserted in the amino acid sequence shown in SEQ ID NO: 5
- examples of the functionally equivalent gene include protein phosphatase 2C gene derived from organisms other than Arabidopsis thaliana.
- Living organisms other than Arabidopsis include rice (Oryza sativa), black cottonwood (Populus trichocarpa), European grapes (Vitis vinifera), tarumago (Medicago truncatula), alfalfa (Medicago sativa), Physcomitrella patens, ice plant (Mesembryanthemum crystallinum), Chlamydomonas reinhardtii, maize (Zea mays), rape (Brassica rapa), tomato (Solanum lycopersicum), mizo physalis (Mimulus guttatus), a single cell red alga Cyanidioschyzon merolae Can be mentioned.
- the gene encoding the glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase can be the At2g01140 gene or a gene functionally equivalent to the gene.
- the gene encoding the glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase preferably encodes one of the following proteins (a) to (c).
- A a protein containing the amino acid sequence shown in SEQ ID NO: 32
- a protein showing fructose 1,6-bisphosphate aldolase activity (c) encoded by a polynucleotide that hybridizes under stringent conditions to a polynucleotide comprising a base sequence complementary to the base sequence shown in SEQ ID NO: 31 And a protein that exhibits fructose 1,6-bisphosphate aldolase activity when glutathione binds.
- the functionally equivalent gene is a glutathione-binding plastid type fructose 1,6 derived from an organism other than Arabidopsis thaliana.
- -Bisphosphate aldolase gene Can be mentioned.
- Organisms other than Arabidopsis are from the group consisting of rice (Oryza sativa), European grape (Vitis vinifera), castor bean (Ricinus communis), black cottonwood (Populus trichocarpa), sitka spruce (Picea sitchensis) and maize (Zea mays) The kind chosen is mentioned.
- Examples of plants targeted in the present invention include dicotyledonous plants such as Arabidopsis plants and Brassicaceae plants.
- the target plant in the present invention includes monocotyledonous plants such as rice and sugar cane among grasses and gramineous plants.
- the plant according to the present invention has significantly improved biomass and / or seeds compared to the wild type.
- the method for increasing the biomass amount and / or the seed amount according to the present invention can significantly increase the biomass amount and / or the seed amount as compared with the wild type of the target plant.
- the method for producing a plant body according to the present invention can produce a plant body in which the biomass amount and / or the seed amount are significantly improved as compared with the wild type. Therefore, by applying the present invention, for example, improvement in productivity when the plant itself is used as a product can be achieved, and cost reduction can be achieved. Further, by applying the present invention, for example, productivity when the seed itself is used as a product and improvement in productivity when a component contained in the seed is used as a product can be achieved. Can be achieved.
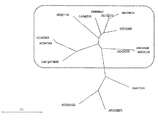
- At1g03590-AtPP2C6-6 At1g16220, At1g79630, At5g01700, At3g02750, At5g36250, At5g26010, At4g32950, At3g16800, At3g05640, At5g27930-AtPP2C6-7, At2g20050 and At3g06270 It is a characteristic view which shows the result of having used the alignment analysis.
- At1g03590-AtPP2C6-6 At1g16220, At1g79630, At5g01700, At3g02750, At5g36250, At5g26010, At4g32950, At3g16800, At3g05640, At5g27930-AtPP2C6-7, At2g20050 and At3g06270 It is a characteristic view which shows the result of having used the alignment analysis.
- At1g03590-AtPP2C6-6 At1g16220, At1g79630, At5g01700, At3g02750, At5g36250, At5g26010, At4g32950, At3g16800, At3g05640, At5g27930-AtPP2C6-7, At2g20050 and At3g06270 It is a characteristic view which shows the result of having used the alignment analysis.
- At1g03590-AtPP2C6-6, At1g16220, At1g79630, At5g01700, At3g02750, At5g36250, At5g26010, At4g32950, At3g16800, At3g05640 and At5g27930-AtPP2C6-7 code sequence using CLUSTAL It is a characteristic view which shows the result.
- At1g03590-AtPP2C6-6, At1g16220, At1g79630, At5g01700, At3g02750, At5g36250, At5g26010, At4g32950, At3g16800, At3g05640 and At5g27930-AtPP2C6-7 code sequence using CLUSTAL It is a characteristic view which shows the result.
- At1g03590-AtPP2C6-6, At1g16220, At1g79630, At5g01700, At3g02750, At5g36250, At5g26010, At4g32950, At3g16800, At3g05640 and At5g27930-AtPP2C6-7 code sequence using CLUSTAL It is a characteristic view which shows the result.
- the FBA1 gene (At2g01140) was transformed into a transformed plant containing an ORF fragment of the PP2C (proteinCphosphatase 2C) gene (At3g05640), or into a transformed plant containing a fragment containing the ORF of the PP2C (protein phosphatase 2C) gene (At3g05640). It is a characteristic figure which shows the result of having measured the biomass amount of the above-ground part of the transformed plant body which introduce
- the plant according to the present invention has a gene encoding a protein phosphatase 2C having a characteristic common sequence and a gene encoding a glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase introduced into the plant, or is endogenous.
- the expression control region of the gene is modified, and the amount of biomass is significantly improved compared to the wild type.
- the plant according to the present invention may be the one obtained by expressing the protein phosphatase 2C gene and the glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene throughout the plant tissue. It may be expressed in at least a part of the tissue.
- the plant tissue is meant to include plant organs such as leaves, stems, seeds, roots and flowers.
- the expression control region means a promoter region to which RNA polymerase binds and a region to which other transcription factors bind.
- the transcription control region it is preferable to replace, for example, the promoter region in the endogenous transcription control region with a promoter region capable of higher expression.
- protein phosphatase 2C gene encodes protein phosphatase 2C having three common sequences consisting of amino acid sequences shown in SEQ ID NOs: 1 to 3 in this order from the N-terminal side.
- This gene encodes protein phosphatase 2C having three common sequences consisting of the amino acid sequences shown in the above from the N-terminal side in this order.
- protein phosphatase 2C genes are predicted in Arabidopsis thaliana, and T-Coffee software (reference: Notredame, C. et al. 2000 T-Coffee: a novel) Method for fast and accurate multiple sequence alignment. J. Mol. Biol. 302, 205-247).
- protein phosphatase 2C genes classified into group E encode protein phosphatase 2C having three common sequences consisting of amino acid sequences shown in SEQ ID NOs: 1 to 3 in this order from the N-terminal side.
- the three common sequences consisting of the amino acid sequences shown in SEQ ID NOs: 1 to 3 are sequences characteristic of group E in the above-described classification, and are sequences that are clearly distinguishable from other groups.
- Group E in the above classification includes At1g03590-AtPP2C6-6, At1g16220, At1g79630, At5g01700, At3g02750, At5g36250, At5g26010, At4g32950, At3g16800, At3g05640, At5g27930-At3g2270 and At2g2270
- the phosphatase 2C gene is included.
- Arabidopsis derived protein phosphatase 2C genes At1g03590-AtPP2C6-6, At1g16220, At1g79630, At5g01700, At3g02750, At5g36250, At5g26010, At4g32950, At3g16800, At3g05640, At5g27930-At3g2 (1.83)
- the results of alignment analysis using multiple sequence alignment program available at DDBJ of National Institute of Genetics (http://clustalw.ddbj.nig.ac.jp/top-j.html)
- the amino acid sequence substitution matrix table used uses the default BLOSUM matrix). As shown in FIG.
- the amino acid residue represented as Xaa is an arbitrary amino acid, and is not limited to any amino acid.
- the first amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 1 is preferably leucine (three letter notation: Leu, one letter notation: L, the same shall apply hereinafter) or phenylalanine (Phe, F).
- the fourth amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 1 is preferably valine (Val, V), isoleucine (Ile, I) or methionine (Met, M).
- the 16th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 1 is preferably serine (Ser, S) or alanine (Ala, A).
- the 17th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 1 is lysine (Lys, K), arginine (Arg, R), glutamine (Gln, Q), or asparagine (Asn, N) Is preferred.
- the common sequence consisting of the amino acid sequence shown in SEQ ID NO: 1 is more specifically (L / F) XG (V / I / M) FDGHGXXGXXX (S / A) (K / R / Q / N) XV Preferably there is.
- this amino acid sequence a plurality of amino acids in parentheses indicate possible amino acid residue variations at that position.
- X means that an arbitrary amino acid residue can be taken at the position.
- this common sequence may be a sequence containing 3 amino acid groups: (D / E / N) XX on the N-terminal side of region I in FIG.
- the amino acid residue represented as Xaa is an arbitrary amino acid and is not limited to any amino acid.
- the fifth amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 2 is preferably glycine (Gly, G), alanine (Ala, A) or serine (Ser, S).
- the sixth amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 2 is preferably valine (Val, V), leucine (Leu, L), or isoleucine (Ile, I).
- the ninth amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 2 is isoleucine (Ile, I), valine (Val, V), phenylalanine (Phe, F), methionine (Met, M) or leucine ( Leu, L) is preferred.
- the 12th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 2 is preferably glycine (Gly, G) or alanine (Ala, A).
- the 15th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 2 is preferably leucine (Leu, L), valine (Val, V), or isoleucine (Ile, I).
- the 17th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 2 is preferably isoleucine (Ile, I), valine (Val, V), or methionine (Met, M).
- the 18th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 2 is preferably glycine (Gly, G) or alanine (Ala, A).
- the 22nd amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 2 is preferably aspartic acid (Asp, D) or histidine (His, H).
- the 26th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 2 is preferably valine (Val, V) or isoleucine (Ile, I).
- the 27th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 2 is preferably leucine (Leu, L), methionine (Met, M), or isoleucine (Ile, I).
- the common sequence consisting of the amino acid sequence shown in SEQ ID NO: 2 is more specifically SGXT (G / A / S) (V / L / I) XX (I / V / F / M / L) XX (G / A) XX (L / V / I) X (I / V / M) (A / G) NXG (D / H) SRA (V / I) (L / M / I)
- a plurality of amino acids in parentheses indicate possible amino acid residue variations at that position.
- X means that an arbitrary amino acid residue can be taken at the position.
- the amino acid residue represented as Xaa is an arbitrary amino acid and is not limited to any amino acid.
- the fourth amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably methionine (Met, M), valine (Val, V), or phenylalanine (Phe, F).
- the fifth amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably serine (Ser, S), alanine (Ala, A), or threonine (Thr, T).
- the seventh amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably alanine (Ala, A) or serine (Ser, S).
- the 8th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably phenylalanine (Phe, F), isoleucine (Ile, I), or valine (Val, V).
- the 14th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably lysine (Lys, K) or glutamic acid (Glu, E).
- the 18th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably valine (Val, V) or leucine (Leu, L).
- the 19th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably isoleucine (Ile, I) or valine (Val, V).
- the 23rd amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably glutamic acid (Glu, E), glutamine (Gln, Q), or aspartic acid (Asp, D).
- the 24th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably isoleucine (Ile, I), valine (Val, V), or phenylalanine (Phe, F).
- the 29th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably isoleucine (Ile, I), leucine (Leu, L), or valine (Val, V).
- the 30th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably serine (Ser, S), threonine (Thr, T), or asparagine (Asn, N).
- the 33rd amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably aspartic acid (Asp, D), asparagine (Asn, N), or histidine (His, H).
- the 35th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably phenylalanine (Phe, F) or tyrosine (Tyr, Y).
- the 36th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is leucine (Leu, L), isoleucine (Ile, I), valine (Val, V), phenylalanine (Phe, F) or methionine ( Met, M) is preferred.
- the 37th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably valine (Val, V), leucine (Leu, L), or isoleucine (Ile, I).
- the 38th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably leucine (Leu, L) or valine (Val, V).
- the 40th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably threonine (Thr, T) or serine (Ser, S).
- the 43rd amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably valine (Val, V), isoleucine (Ile, I) or methionine (Met, M).
- the 44th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably tryptophan (Trp, W) or phenylalanine (Phe, F).
- the 45th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably aspartic acid (Asp, D) or glutamic acid (Glu, E).
- the 47th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably leucine (Leu, L), isoleucine (Ile, I), or methionine (Met, M).
- the 48th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably serine (Ser, S), threonine (Thr, T), or proline (Pro, P).
- the 49th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably asparagine (Asn, N) or serine (Ser, S).
- the 52nd amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably valine (Val, V) or alanine (Ala, A).
- the 55th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is leucine (Leu, L), valine (Val, V), isoleucine (Ile, I) or methionine (Met, M) Is preferred.
- the 56th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is preferably isoleucine (Ile, I) or valine (Val, V). That is, as a common sequence consisting of the amino acid sequence shown in SEQ ID NO: 3, more specifically, GXA (M / V / F) (S / A / T) R (A / S) (F / I / V) GDXXX ( K / E) XXG (V / L) (I / V) XXP (E / Q / D) (I / V / F) XXXX (I / L / V) (T / S) XX (D / N / H ) X (F / Y) (L / I / V / F) (V / L / I) (L / V) A (T / S) DG (V / I / M) (W / F) (
- the 20th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is more preferably alanine (Ala, A), serine (Ser, S), or cysteine (Cys, C).
- the 50th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 3 is aspartic acid (Asp, D), glutamic acid (Glu, E), lysine (Lys, K), glutamine (Gln, Q). Or asparagine (Asn, N) is more preferable.
- reference (2) Henikoff S., Henikoff JG, Amino-acid substitution matrices from protein blocks, Proc. Natl. Acad.Sci. USA, USA89, 10915-10919 (1992), Fig. A score matrix (BLOSUM) for substitution mutations of amino acid residues was proposed and widely used in .2.
- amino acid substitution between similar side chain chemical properties is based on the knowledge that structural and functional changes given to the whole protein are reduced.
- the side chain groups of amino acids considered in multiple alignment can be considered based on indicators such as chemical properties and physical size.
- score matrix (BLOSUM) disclosed in reference (2) as an amino acid group with a score of 0 or more, preferably a group of amino acids with a value of 1 or more.
- the following eight groups are listed as typical groups.
- Other fine groupings may be any amino acid group of 0 or more, preferably 1 or more, more preferably 2 or more amino acid groups of the score value.
- Aliphatic hydrophobic amino acid group This group is a group of amino acids having a hydrophobic hydrophobic side chain among the neutral non-polar amino acids shown in the above-mentioned reference (1), V (Val, valine), L (Leu, leucine) , I (Ile, isoleucine) and M (Met, methionine).
- V Val, valine
- L Leu, leucine
- I Ile, isoleucine
- M Metal, methionine
- FGACWP is not included in this “aliphatic hydrophobic amino acid group” for the following reasons. This is because G (Gly, glycine) and A (Ala, alanine) are less than a methyl group and have a weak nonpolar effect.
- C Cys, cysteine
- F Phenylalanine
- W Trp, tryptophan
- P Pro, proline
- ST group Group with hydroxymethylene group
- S Ser, serine
- T Thr, threonine
- Acidic amino acids This group is a group of amino acids having an acidic carboxyl group in the side chain, and is composed of D (Asp, aspartic acid) and E (Glu, glutamic acid).
- KR group This group is a group of basic amino acids and is composed of K (Lys, lysine) and R (Arg, arginine). These K and R are positively charged and have basic properties over a wide pH range. On the other hand, H (His, histidine) classified as a basic amino acid is not classified into this group because it is hardly ionized at pH 7.
- Methylene group polar group (DHN group) This group has a feature that a methylene group is bonded as a side chain to a carbon element at the ⁇ -position and has a polar group at the tip.
- Dimethylene group polar group (EKQR group) This group has a feature that a linear hydrocarbon having a dimethylene group or more is bonded as a side chain to a carbon element at the ⁇ -position and has a polar group at the tip.
- E Glu, glutamic acid, polar group is carboxyl group
- K Lis, lysine, polar group is amino group
- Q Gln, glutamine, polar group is amide group
- R Arg, arginine, polar group is imino group
- Aromatic (FYW Group) This group is an aromatic amino acid with a benzene nucleus in the side chain and is characterized by aromatic chemical properties. It consists of F (Phe, phenylalanine), Y (Tyr, tyrosine), W (Trp, tryptophan).
- Circular & polar (HY group) This group is an amino acid that has a cyclic structure in the side chain and also a polarity, H (H, histidine, both cyclic structure and polar group are imidazole groups), Y (Tyr, tyrosine, cyclic structure is polar with benzene nucleus The group consists of hydroxyl).
- the amino acid residue shown as Xaa may be any amino acid, but the amino acid residue shown as Xaa is within the group of 1) to 8) above. It can be seen that amino acid substitution may be performed. That is, in the present invention, the protein phosphatase 2C gene that is introduced into the plant body or modifies the expression control region has three common sequences consisting of amino acid sequences shown in SEQ ID NOs: 1 to 3 in this order from the N-terminal side. Any plant-derived protein phosphatase 2C gene may be used.
- genes encoding protein phosphatase 2C having three common sequences consisting of amino acid sequences shown in SEQ ID NOs: 1 to 3 in this order from the N-terminal side in Arabidopsis thaliana are At1g03590-AtPP2C6-6, At1g16220, At1g79630 At5g01700, At3g02750, At5g36250, At5g26010, At4g32950, At3g16800, At3g05640, At5g27930-AtPP2C6-7, At2g20050 and At3g06270, in the present invention, at least one gene selected from these gene groups is introduced into the plant body, Alter the expression control region of the gene of interest.
- At1g03590-AtPP2C6-6 At1g16220, At1g79630, At5g01700, At3g02750, At5g36250, At5g26010, At4g32950, At3g16800, At3g05640 and At5g27930-AtPP2C6-7 is introduced.
- At3g16800, At3g05640 and At5g27930-AtPP2C6-7 it is more preferable to introduce at least one gene selected from At3g16800, At3g05640 and At5g27930-AtPP2C6-7 into a plant body, or to modify the expression control region of the gene concerned, Most preferably, the gene to be identified is introduced into the plant body or the expression control region of the gene concerned is altered.
- FIG. 2 shows three common protein phosphatases encoded by At1g03590-AtPP2C6-6, At1g16220, At1g79630, At5g01700, At3g02750, At5g36250, At5g26010, At4g32950, At3g16800, At3g05640, and At5g27930-AtPP2C6-7. Yes.
- FIG. 2 shows three common protein phosphatases encoded by At1g03590-AtPP2C6-6, At1g16220, At1g79630, At5g01700, At3g02750, At5g36250, At5g26010, At4g32950, At3g16800, At3g05640, and At5g27930-AtPP2C6-7.
- the common sequence shown in SEQ ID NO: 48 is (L / F) CG (V / I / M) FDGHGXXGXX (V / I) (S / A) (K / R) XV. More specifically, the consensus sequence shown in SEQ ID NO: 49 is SGXT (G / A / S) (V / L) XX (I / V / F / L) XX (G / A) XX (L / V / I) X (I / V / M) (A / G) NXG (D / H) SRA (V / I) (L / M / I).
- SEQ ID NO: 50 is GLA (M / V) (S / A) R (A / S) (F / L) GDXX (L / I / V) KX (Y / F / H ) G (V / L) (I / V) XXP (E / Q / D) (I / V / F) XXXX (I / L / V) (T / S) XXDX (F / Y) (L / I / V / M) (V / L / I) LA (T / S) DG (V / I / M) WDX (L / I / M / V) (S / T) NX (E / D) (V / A) XX (L / V / I) (I / V).
- amino acid sequences a plurality of amino acids in parentheses indicate possible amino acid residue variations at that position.
- X means that an arbitrary amino acid residue can be taken at the position.
- the ninth amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 49 is more preferably isoleucine (Ile, I), valine (Val, V), or phenylalanine (Phe, F).
- the 11th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 49 is more preferably glutamine (Gln, Q) or histidine (His, H).
- the 13th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 49 is lysine (Lys, K), glutamic acid (Glu, E), serine (Ser, S), glutamine (Gln, Q), asparagine More preferably, it is an acid (Asp, D) or asparagine (Asn, N).
- the seventh amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 50 is more preferably alanine (Ala, A).
- the 8th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 50 is more preferably phenylalanine (Phe, F).
- the 11th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 50 is more preferably phenylalanine (Phe, F) or tyrosine (Tyr, Y).
- the 13th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 50 is more preferably leucine (Leu, L) or isoleucine (Ile, I).
- the 15th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 50 is more preferably aspartic acid (Asp, D), serine (Ser, S), or glutamic acid (Glu, E).
- the 20th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 50 is more preferably serine (Ser, S), alanine (Ala, A), or cysteine (Cys, C).
- the 27th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 50 is more preferably histidine (His, H) or arginine (Arg, R).
- the 34th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 50 is more preferably glutamine (Gln, Q), glutamic acid (Glu, E), or histidine (His, H).
- the 36th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 50 is more preferably leucine (Leu, L), isoleucine (Ile, I) or valine (Val, V).
- the 47th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 50 is more preferably leucine (Leu, L), isoleucine (Ile, I) or valine (Val, V).
- the 50th amino acid residue from the N-terminal side in the amino acid sequence shown in SEQ ID NO: 50 is lysine (Lys, K), glutamic acid (Glu, E), glutamine (Gln, Q), aspartic acid (Asp, D). Or asparagine (Asn, N) is more preferable.
- the base sequence of the coding region in the gene specified by At3g05640 is shown in SEQ ID NO: 4, and the amino acid sequence of protein phosphatase 2C encoded by the gene specified by At3g05640 is shown in SEQ ID NO: 5.
- genes functionally equivalent to the genes listed above may be introduced into the plant body, or the expression control region of the gene concerned may be modified.
- the functionally equivalent gene refers to, for example, three common sequences derived from organisms other than Arabidopsis thaliana and consisting of amino acid sequences shown in SEQ ID NOs: 1 to 3 (preferably 3 consisting of amino acid sequences shown in SEQ ID NOs: 48 to 50). It includes the gene encoding protein phosphatase 2C having two common sequences (the same applies hereinafter) in this order from the N-terminal side.
- a functionally equivalent gene means a gene encoding a protein having protein phosphatase 2C activity.
- Protein phosphatase 2C activity means Mg 2+ or Mn 2+ dependent serine / threonine phosphatase (Ser / Thr phosphatase) activity. Therefore, whether a gene encodes a protein having protein phosphatase 2C activity can be determined by examining whether the product of the gene has serine / threonine phosphatase activity in the presence of Mg 2+ or Mn 2+ .
- a method for measuring serine / threonine phosphatase activity a conventionally known method can be appropriately used. For example, a commercially available activity measurement kit ProFluor (registered trademark) Ser / Thr Phosphatase Assay (manufactured by Promega) can be used.
- examples of organisms other than Arabidopsis include, but are not limited to, for example, rice. That is, examples of functionally equivalent genes include the Os05g0358500 gene in rice. The base sequence of the coding region of the Os05g0358500 gene is shown in SEQ ID NO: 6, and the amino acid sequence of the protein encoded by the gene is shown in SEQ ID NO: 7.
- rice-derived genes having the above functional equivalents include Os11g0109000 (base sequences and amino acid sequences are shown in SEQ ID NOs: 8 and 9), Os12g0108600 (base sequences and amino acid sequences are shown in SEQ ID NOs: 10 and 11).
- Os02g0471500 base sequence and amino acid sequence are shown in SEQ ID NOs: 12 and 13
- Os04g0321800 base sequence and amino acid sequence are shown in SEQ ID NOs: 14 and 15
- Os11g0417400 base sequence and amino acid sequence are shown in SEQ ID NO: 16 and 17
- Os07g0566200 base sequences and amino acid sequences are shown in SEQ ID NOs: 18 and 19
- Os08g0500300 base sequences and amino acid sequences are shown in SEQ ID NOs: 20 and 21
- Os02g0224100 base sequences and amino acid sequences are shown in SEQ ID NO: 22
- Os02g0281000 base sequences and amino acid sequences are shown in SEQ ID NOs: 51 and 52).
- genes derived from plants other than Arabidopsis and rice, and the functionally equivalent genes include genes derived from black cottonwood (Populus trichocarpa) (UniProt database accession numbers A9P973, A9PFS0 and A9P7U4).
- genes from European grape (Vitis vinifera) (UniProt database accession numbers A7PRZ8, A7Q8H4, A7PV59, A5C3B0, A5BF43, A7QFG6, A7P4H7, A5C0C9, A5AP53, A7QQF9 and A5BDPgo ) Genes (UniProt database accession numbers Q2HW33 and Q4L0F8), genes derived from alfalfa (Medicago sativa) (GenBank database accession number AY651248), and genes derived from Physcomitrella patens (UniPr ot database accession numbers A9SE70, A9SE69 and A9RFU1), genes derived from ice plant (Mesembryanthemum crystallinum) (UniProt database accession number 2511453C), and genes derived from Chlamydomonas reinhardtii (Chlamydomonas reinhardtii (Ch
- the genes encoding protein phosphatase 2C having three common sequences consisting of the amino acid sequences shown in SEQ ID NOs: 1 to 3 in this order from the N-terminal side in plants other than Arabidopsis thaliana represented by these are derived from Arabidopsis thaliana listed above. Based on the base sequence of the protein phosphatase 2C gene and the amino acid sequence of protein phosphatase 2C, it can be easily searched and identified from known databases such as GenBank.
- the protein phosphatase 2C gene is not limited to the protein phosphatase 2C gene comprising the base sequence and amino acid sequence shown in SEQ ID NOs: 4 to 23 as described above. That is, the protein phosphatase 2C gene includes an amino acid sequence in which one or a plurality of amino acid sequences are deleted, substituted, added or inserted in the amino acid sequences shown in odd numbers of SEQ ID NOs: 4 to 23, and protein phosphatase It may have 2C activity.
- the plurality of amino acids for example, 1 to 20, preferably 1 to 10, more preferably 1 to 7, further preferably 1 to 5, particularly preferably 1 to 3 are used. means.
- amino acid deletion, substitution, or addition can be performed by modifying the base sequence encoding the protein phosphatase 2C gene by a technique known in the art.
- Mutation can be introduced into a nucleotide sequence by a known method such as Kunkel method or Gapped duplex method or a method according thereto, for example, a mutation introduction kit using site-directed mutagenesis (for example, Mutant- Mutations are introduced using K, Mutant-G (both trade names, manufactured by TAKARA Bio Inc.) or the like, or using LA PCR-in-vitro Mutagenesis series kits (trade name, manufactured by TAKARA Bio Inc.).
- EMS ethyl methanesulfonic acid
- 5-bromouracil 2-aminopurine
- hydroxylamine N-methyl-N'-nitro-N nitrosoguanidine
- other carcinogenic compounds are representative.
- a method using a chemical mutagen such as that described above may be used, or a method using radiation treatment or ultraviolet treatment represented by X-rays, alpha rays, beta rays, gamma rays and ion beams may be used.
- the protein phosphatase 2C gene may be a homologous gene of the protein phosphatase 2C gene consisting of the base sequence and amino acid sequence shown in SEQ ID NOs: 4 to 23.
- the homologous gene generally means a gene that has evolved and branched from a common ancestral gene, and is generated by two kinds of homologous genes (ortholog) and duplicated branching within the same species. It means to include homologous genes (paralog).
- the above-mentioned “functionally equivalent gene” includes homologous genes such as orthologs and paralogs.
- the above-mentioned “functionally equivalent genes” include genes that do not evolve from a common gene but simply have similar functions.
- the similarity to these amino acid sequences is 70% or more, preferably 80% or more, more preferably A protein having protein phosphatase 2C activity having an amino acid sequence of 90% or more, most preferably 95% or more, and having three common sequences consisting of the amino acid sequences shown in SEQ ID NOs: 1 to 3 in this order from the N-terminal side Mention may be made of the genes encoded.
- the similarity value means a value obtained by default setting using a computer program in which a BLAST (Basic Local Alignment Search Tool) program is implemented and a database storing gene sequence information.
- a genome is constructed that constructs a cDNA library of the target plant and hybridizes under stringent conditions to at least part of the protein phosphatase 2C gene consisting of the nucleotide sequence and amino acid sequence shown in SEQ ID NOs: 4 to 23. It can be identified by isolating the region or cDNA.
- stringent conditions refer to conditions under which so-called specific hybrids are formed and non-specific hybrids are not formed. For example, hybridization at 45 ° C.
- Hybridization can be performed by a conventionally known method such as the method described in J. Sambrook et al. OleMolecular lonCloning, A Laboratory Manual, 2nd Ed., Cold Spring Harbor Laboratory (1989).
- a method for introducing this protein phosphatase 2C gene into a plant a method for introducing an expression vector in which an exogenous protein phosphatase 2C gene is arranged under the control of a promoter that enables expression in the plant can be mentioned.
- Examples of the technique for modifying the expression control region of the endogenous gene include a technique for modifying the promoter of the endogenous protein phosphatase 2C gene in the target plant body.
- a method of introducing an expression vector in which the above-described protein phosphatase 2C gene is placed under the control of a promoter capable of expression in a plant body is preferable.
- Glutathione-binding plastid fructose 1,6-bisphosphate aldolase gene the activity of the glutathione-binding plastid fructose 1,6-bisphosphate aldolase gene is regulated by binding to glutathione, such as chloroplasts, etc. It is a gene encoding an enzyme having an activity of catalyzing a reaction (reversible reaction) for converting fructose 1,6-bisphosphate into dihydroxyacetone phosphate and glyceraldehyde 3-phosphate in plastid.
- the glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene may be any plant-derived gene as long as it encodes an enzyme having the above-mentioned activity, or a gene isolated from a plant is mutated. It may also be a mutant gene into which is introduced.
- a glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene derived from Arabidopsis thaliana can be used.
- a glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene derived from Arabidopsis thaliana is disclosed as an FBA1 gene in WO2007 / 091634.
- the base sequence of the coding region in this FBA1 gene is shown in SEQ ID NO: 31, and the amino acid sequence of glutathione-binding plastid fructose 1,6-bisphosphate aldolase encoded by the FBA1 gene is shown in SEQ ID NO: 32.
- the FBA1 gene in Arabidopsis is also referred to as the At2g01140 gene.
- a gene functionally equivalent to the above At2g01140 gene may be introduced into the plant body, or the expression control region of the gene concerned may be modified.
- the functionally equivalent gene is meant to include, for example, genes derived from organisms other than Arabidopsis thaliana and having glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase activity.
- the fructose 1,6-bisphosphate aldolase activity is obtained by, for example, reacting a protein to be measured with a buffer containing fructose 1,6-bisphosphate as a substrate, and producing dihydroxyacetone phosphate and / or glyceraldehyde- It can be measured by measuring 3-phosphate.
- fructose 1,6-bisphosphate aldolase activity can also be measured as follows. That is, first, the protein whose activity is to be measured is allowed to act in a buffer containing fructose 1,6-bisphosphate as a substrate. Triosephosphate isomerase is allowed to act on the glyceraldehyde-3-phosphate produced to produce dihydroxyacetone phosphate. When this dihydroxyacetone phosphate is changed to glycerol-3-phosphate in the presence of glycerol-3-phosphate dehydrogenase, NADH becomes ⁇ -nicotinamide adenine dinucleotide oxidized form (NAD). The resulting absorbance at 340 nm decreases. Therefore, the aldolase activity of the protein whose activity is to be measured can be evaluated by measuring the rate of decrease of NADH.
- Triosephosphate isomerase is allowed to act on the glyceraldehyde-3-phosphate produced to produce dihydroxyacetone phosphate.
- the glutathione binding property of the protein to be evaluated can be evaluated by the presence or absence of a glutathione binding sequence in the amino acid sequence of the protein, or the protein to be evaluated is allowed to act in the presence of glutathione transferase and glutathione to bind glutathione. It can also be evaluated by measuring the presence or absence.
- whether the protein to be evaluated is a plastid type can be evaluated by the presence or absence of a transit peptide sequence in the amino acid sequence of the protein, or a gene encoding a fusion protein of the protein and a reporter such as a fluorescent protein. It is also possible to evaluate by detecting a plastid localization of the protein by producing a plant into which is introduced and detecting a reporter.
- organisms other than Arabidopsis thaliana are not limited in any way, and examples thereof include rice (Oryza sativa). That is, examples of functionally equivalent genes include the Os01g0118000 gene in rice.
- the amino acid sequence of the protein encoded by the Os01g0118000 gene is shown in SEQ ID NO: 43.
- genes derived from plants other than Arabidopsis and rice and having the above functional equivalents include genes derived from European grapevine (Vitis vinifera) (NCBI Entrez Protein database accession number CAO42215 (SEQ ID NO: 40)), castor bean (Ricinus communis) gene (NCBI Entrez Protein database accession number EEF36097 (SEQ ID NO: 39)), black cottonwood (Populus trichocarpa) gene (NCBI Entrez Protein database accession number EEE88847 (SEQ ID NO: 38) EEF02079 (SEQ ID NO: 36) and ABK94899 (SEQ ID NO: 37)), genes derived from Sitka spruce (Piceaicesitchensis) (NCBI Entrez Protein database accession numbers ABK24568 (SEQ ID NO: 35), ABK24286 (SEQ ID NO: 33) and ABK25226 ( SEQ ID NO: 34)) and remains from maize (Zea mays) Can be given child
- Genes having glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase activity in plants other than those represented by Arabidopsis are represented by the base sequence of the FBA1 gene (At2g01140 gene) derived from Arabidopsis thaliana listed above and the gene concerned. Based on the amino acid sequence of the encoded protein, it can be easily searched and identified from a known database such as GenBank.
- the CLUSTAL W (1.83) multiple sequence alignment program (available at the National Institute of Genetics DDBJ (http://clustalw.ddbj.nig.ac.jp/top-j .html)) is shown in FIG. 3 (the amino acid sequence substitution matrix table used is a default BLOSUM matrix).
- FIG. 3 it can be seen that the glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene shows very high homology.
- the glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene having the amino acid sequence shown in SEQ ID NOs: 32-43 and the fructose 1,6-bisphosphate aldolase gene (FBA2 gene (At4g38970), FBA3 gene (At2g21330) in Arabidopsis thaliana) )
- FBA2 gene At4g38970
- FBA3 gene At2g21330 in Arabidopsis thaliana
- the glutathione-binding plastid fructose 1,6-bisphosphate aldolase gene having the amino acid sequence shown in SEQ ID NOs: 32 to 43 includes the FBA2 gene in Arabidopsis, the FBA3 gene, and the BAA77604 gene in tobacco. Form a different group.
- fructose 1,6-bisphosphate aldolase encoded by the FBA2 gene and the FBA3 gene in Arabidopsis thaliana does not have the characteristic of exhibiting fructose 1,6-bisphosphate aldolase activity by glutathione.
- the glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene encodes a protein comprising the base sequence shown in SEQ ID NO: 31 and the amino acid sequences shown in SEQ ID NOs: 32-43 as described above. It is not limited to the gene to be. That is, the glutathione-binding plastid type fructose 1,6-bisphosphate aldolase gene includes an amino acid sequence in which one or a plurality of amino acid sequences are deleted, substituted, added or inserted in the amino acid sequences shown in SEQ ID NOs: 32-43. And having glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase activity.
- the plurality of amino acids for example, 1 to 20, preferably 1 to 10, more preferably 1 to 7, further preferably 1 to 5, particularly preferably 1 to 3 are used. means.
- the method disclosed in the column of “Protein Phosphatase 2C gene” described above can be applied.
- the glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene is a homologous gene of a gene encoding a protein consisting of the nucleotide sequence shown in SEQ ID NO: 31 and the amino acid sequence shown in SEQ ID NOs: 32-43. May be.
- the homologous gene generally means a gene that has evolved and branched from a common ancestral gene, and is generated by two kinds of homologous genes (ortholog) and duplicated branching within the same species. It means to include homologous genes (paralog).
- the above-mentioned “functionally equivalent gene” includes homologous genes such as orthologs and paralogs.
- the above-mentioned “functionally equivalent genes” include genes that do not evolve from a common gene but simply have similar functions.
- the similarity to these amino acid sequences is, for example, 70% or more, preferably A gene encoding a protein having an amino acid sequence of 80% or more, more preferably 90% or more, most preferably 95% or more, and having glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase activity. it can.
- the value of similarity means a value to which the technique disclosed in the column of “protein phosphatase 2C gene” described above is applied.
- the gene containing the base sequence shown in SEQ ID NO: 31 and the gene similar to the gene encoding the protein consisting of the amino acid sequence shown in SEQ ID NO: 32 to 43 when plant genome information is not clear, Extract the genome from the target plant or construct a cDNA library of the target plant and code the gene comprising the base sequence shown in SEQ ID NO: 31 and the protein consisting of the amino acid sequence shown in SEQ ID NOs: 32-43 It can be identified by isolating a genomic region or cDNA that hybridizes under stringent conditions to at least a part of the gene.
- the stringent condition means the condition disclosed in the column of “protein phosphatase 2C gene” described above.
- the technique disclosed in the column of “Protein Phosphatase 2C Gene” described above can be applied.
- the expression vector expression vector is constructed so as to contain a promoter capable of expression in a plant, the protein phosphatase 2C gene and the glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene described above.
- An expression vector containing the protein phosphatase 2C gene and an expression vector containing a glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene may be prepared separately.
- a plasmid, phage, cosmid or the like can be used, and can be appropriately selected according to the plant cell to be introduced and the introduction method.
- Specific examples include pBR322, pBR325, pUC19, pUC119, pBluescript, pBluescriptSK, and pBI vectors.
- the method for introducing a vector into a plant is a method using Agrobacterium, it is preferable to use a pBI binary vector.
- the pBI binary vector include pBIG, pBIN19, pBI101, pBI121, pBI221, and the like.
- the promoter is not particularly limited as long as it is a promoter capable of expressing the protein phosphatase 2C gene and glutathione-binding plastid fructose 1,6-bisphosphate aldolase gene in plants, and a known promoter is preferably used.
- a known promoter is preferably used.
- Such promoters include cauliflower mosaic virus 35S promoter (CaMV35S), various actin gene promoters, various ubiquitin gene promoters, nopaline synthase gene promoter, tobacco PR1a gene promoter, tomato ribulose 1,5-diphosphate carboxylase Oxidase small subunit gene promoter, napin gene promoter, etc.
- cauliflower mosaic virus 35S promoter, actin gene promoter, or ubiquitin gene promoter can be more preferably used. When each of the above promoters is used, any gene can be strongly expressed when introduced into a plant cell.
- a promoter having a function of expressing in a site-specific manner in a plant can be used.
- any conventionally known promoter can be used.
- the expressed plant organ is compared with the wild type. Can be increased.
- the expression vector may further contain other DNA segments in addition to the promoter, the protein phosphatase 2C gene and the glutathione-binding plastid fructose 1,6-bisphosphate aldolase gene.
- the other DNA segment is not particularly limited, and examples thereof include a terminator, a selection marker, an enhancer, and a base sequence for improving translation efficiency.
- the recombinant expression vector may further have a T-DNA region.
- the T-DNA region can increase the efficiency of gene transfer particularly when Agrobacterium is used to introduce the recombinant expression vector into a plant body.
- the transcription terminator is not particularly limited as long as it has a function as a transcription termination site, and may be a known one.
- the transcription termination region (Nos terminator) of the nopaline synthase gene the transcription termination region of the cauliflower mosaic virus 35S (CaMV35S terminator) and the like can be preferably used.
- the Nos terminator can be more preferably used.
- a transcription terminator is placed at an appropriate position, so that after introduction into a plant cell, an unnecessarily long transcript is synthesized, and a strong promoter reduces the copy number of the transcript. Occurrence of the phenomenon can be prevented.
- a transformant selection marker for example, a drug resistance gene can be used.
- drug resistance genes include drug resistance genes for hygromycin, bleomycin, kanamycin, gentamicin, chloramphenicol and the like.
- Examples of the base sequence for improving the translation efficiency include an omega sequence derived from tobacco mosaic virus. By placing this omega sequence in the untranslated region (5′UTR) of the promoter, the translation efficiency of the fusion gene can be increased.
- the recombinant expression vector can contain various DNA segments depending on the purpose.
- the method for constructing the recombinant expression vector is not particularly limited, and the promoter, the protein phosphatase 2C gene and / or the glutathione-binding plastid-type fructose 1,6-bislin can be selected as a mother vector appropriately selected. What is necessary is just to introduce
- an expression cassette is constructed by linking the protein phosphatase 2C gene or the glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene and a promoter (such as a transcription terminator if necessary), and using this as a vector What is necessary is just to introduce.
- a promoter such as a transcription terminator if necessary
- the order of the DNA segments can be defined by setting the cleavage sites of each DNA segment as complementary protruding ends and reacting with a ligation enzyme.
- the promoter, the protein phosphatase 2C gene or the glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene, and the terminator may be in order from the upstream.
- the types of reagents for constructing the expression vector that is, the types of restriction enzymes and ligation enzymes are not particularly limited, and commercially available ones may be appropriately selected and used.
- the method for producing the above expression vector is not particularly limited, and a conventionally known method can be used.
- Escherichia coli may be used as a host and propagated in the E. coli.
- a preferred E. coli type may be selected according to the type of vector.
- the expression vector described above is introduced into a target plant by a general transformation method.
- the method (transformation method) for introducing the expression vector into the plant cell is not particularly limited, and any conventionally known method suitable for the plant cell can be used. Specifically, for example, a method using Agrobacterium or a method of directly introducing into plant cells can be used.
- a method using Agrobacterium for example, Bechtold, E., Ellis, J. and Pelletier, G. (1993) In Planta Agrobacterium-mediated gene transfer by infiltration of adult Arabidopsis plants. CR Acad. Sci. Paris Sci. Vie, 316, 1194-1199.
- a method for directly introducing an expression vector into a plant cell for example, a microinjection method, an electroporation method (electroporation method), a polyethylene glycol method, a particle gun method, a protoplast fusion method, a calcium phosphate method, or the like can be used.
- a transcription unit necessary for expression of the target gene such as a promoter or transcription terminator, and DNA containing the target gene. Function is not essential. Furthermore, even if it is DNA which contains only the protein coding region of the gene of interest which does not have a transcription unit, it can be integrated into the transcription unit of the host to express the gene of interest.
- Examples of the plant cell into which the expression vector or the expression cassette containing the gene of interest without the expression vector is introduced include, for example, cells of each tissue in plant organs such as flowers, leaves, roots, callus, suspension culture, etc. A cell etc. can be mentioned.
- the expression vector may be appropriately constructed according to the type of plant to be produced, but a general-purpose expression vector is constructed in advance and introduced into plant cells. Also good.
- the plant to which the expression vector is introduced in other words, the plant to be increased in biomass production is not particularly limited. That is, by introducing the protein phosphatase 2C gene and the glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene described above, an effect of increasing biomass production can be expected for all plants.
- Examples of the target plant include dicotyledonous plants and monocotyledonous plants, for example, plants belonging to the family Brassicaceae, Gramineae, Eggplant, Legume, Willow, etc. (see below), but are limited to these plants. Is not to be done.
- Brassicaceae Arabidopsis thaliana, rape (Brassica rapa, Brassica napus, Brassica campestris), cabbage (Brassica oleracea var. Capitata), Chinese cabbage (Brassica rapa var. Pekinensis), chinensai (Brassica Brassica rapa var. Rapa), Nozawana (Brassica rapa var. (Wasabia japonica).
- Eggplant family tobacco (Nicotiana tabacum), eggplant (Solanum melongena), potato (Solaneum tuberosum), tomato (Lycopersicon lycopersicum), capsicum (Capsicum annuum), petunia (Petunia), etc.
- Legumes Soybean (Glycine max), peas (Pisum sativum), broad bean (Vicia faba), wisteria (Wisteria floribunda), groundnut (Arachis hypogaea), Miyakogusa (Lotus corniculatus var. Japonicus), common bean (garten) Vigna angularis), Acacia, etc.
- Asteraceae chrysanthemum (Chrysanthemum morifolium), sunflower (Helianthus annuus), etc.
- Palms oil palm (Elaeis guineensis, Elaeis oleifera), coconut (Cocos nucifera), date palm (Phoenix dactylifera), wax palm (Copernicia), etc.
- Ursiaceae Hazenoki (Rhus succedanea), Cashewnut (Anacardium occidentale), Urushi (Toxicodendron vernicifluum), Mango (Mangifera indica), Pistachio (Pistacia vera), etc.
- Cucurbitaceae pumpkins (Cucurbita maxima, Cucurbita moschata, Cucurbita pepo), cucumbers (Cucumis sativus), callas (Trichosanthes cucumeroides), gourds (Lagenaria siceraria var. Gourda), etc.
- Rosaceae Almond (Amygdalus communis), Rose (Rosa), Strawberry (Fragaria), Sakura (Prunus), Apple (Malus pumila var. Domestica).
- Dianthus Carnation (Dianthus caryophyllus).
- Gramineae maize (Zea mays), rice (Oryza sativa), barley (Hordeum vulgare), wheat (Triticum aestivum), bamboo (Phyllostachys), sugar cane (Saccharum officinarum), napiergrass (Pennisetum pupureum) ), Miscanthus virgatum, Sorghum switchgrass (Panicum), etc.
- Lily family Tulip (Tulipa), Lily (Lilium), etc.
- biofuels such as sugarcane, corn, rapeseed, and sunflower.
- biofuels such as sugarcane, corn, rapeseed, and sunflower.
- biofuels such as bioethanol, biodiesel, biomethanol, bio DME, bio GTL (BTL) and biobutanol by increasing the biomass of energy crops.
- the protein phosphatase 2C gene that can be used in the present invention and the glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene can be isolated from various plants and used. Depending on the type of plant to be increased, it can be appropriately selected and used.
- the biomass production target plant is a monocotyledonous plant
- a protein phosphatase 2C gene isolated from the monocotyledonous plant when the plant whose biomass production is to be increased is rice, it is preferable to introduce a rice-derived protein phosphatase 2C gene (SEQ ID NO: 6).
- the glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene can be selected according to the plant whose biomass is to be increased.
- a protein phosphatase 2C gene derived from a dicotyledonous plant or a glutathione-binding plastid fructose 1,6-bisphosphate aldolase gene is introduced. Also good. That is, for example, Arabidopsis derived protein phosphatase 2C gene (SEQ ID NO: 4) and glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene (SEQ ID NO: 31) are not limited to dicotyledonous plants but are widely used in monocotyledonous plants. It may be introduced to be expressed in the plant to be classified.
- an expression vector containing a protein phosphatase 2C gene and an expression vector containing a glutathione-binding plastid fructose 1,6-bisphosphate aldolase gene are prepared separately, these expression vectors are used together for transformation.
- the transformed plant may be further transformed with the other expression vector.
- the self-propagating seed of the transformed plant is collected, and after confirming that the gene introduced in the progeny plant is fixed, On the other hand, it can be transformed with the other expression vector.
- a transformed plant into which the protein phosphatase 2C gene was introduced and a transformed plant into which the glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene was introduced were crossed.
- a progeny plant having both a protein phosphatase 2C gene and a glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene may be obtained.
- the plant body in which the expression of protein phosphatase 2C gene and glutathione-binding plastid fructose 1,6-bisphosphate aldolase gene, which are inherently possessed by the plant body, is activated by induction of mutation, introduction of gene activator, etc. May be selected.
- a selection step for selecting an appropriate transformant from among plant bodies can be performed by a conventionally known method.
- the selection method is not particularly limited.
- the selection may be performed on the basis of drug resistance such as hygromycin resistance, and after growing the transformant, the plant itself, or any organ or tissue You may select the thing which measured weight and has increased significantly compared with the wild type.
- progeny plants can be obtained from the transformed plants obtained by the transformation treatment according to a conventional method.
- a biomass amount of a progeny plant having a trait introduced with the protein phosphatase 2C gene and the glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene or a trait in which the expression control region of the gene is modified By selecting on the basis of the above, it is possible to produce a stable plant line in which the biomass amount is increased by having the above traits.
- plant cells, seeds, fruits, strains, callus, tubers, cuttings, clumps, and other propagation materials are obtained from the transformed plants and their progeny, and the amount of biomass is increased by having the above traits based on these. It is also possible to mass-produce stable plant lines.
- the plant body in the present invention includes at least one of a grown plant individual, a plant cell, a plant tissue, a callus, and a seed. That is, in this invention, if it is a state which can be made to grow finally to a plant individual, all will be considered as a plant body.
- the plant cells include various forms of plant cells. Such plant cells include, for example, suspension culture cells, protoplasts, leaf sections and the like. Plants can be obtained by growing and differentiating these plant cells. In addition, the reproduction
- the above-described protein phosphatase 2C gene and the glutathione-binding plastid fructose 1,6-bisphosphate aldolase gene are introduced into, or the expression control region of the endogenous gene is modified.
- the significant increase in the biomass amount means that the total weight per individual is statistically significantly larger than that of the wild type. At this time, even if some tissues of the plant body were specifically large and other tissues were equivalent to the wild type, if the total weight of the whole plant body was large, it was judged that the biomass amount increased. To do.
- a significant increase in the amount of seeds means that the total amount and / or total number of seeds collected from one individual is statistically significantly larger than that of the wild type. That is, when the size per seed is improved, when the size per seed is the same and the number of seeds is improved, or the size per seed is improved, and the number of seeds is Any case of improvement is possible.
- the biomass amount and / or seed amount of the plant body is increased, when the whole plant body is intended for production, a part of the plant body (such as seeds) and its components are produced. In any case, improvement in productivity can be achieved.
- the fats and oils contained in plant seeds are intended for production, the amount of fats and oils that can be recovered per planted area can be greatly improved.
- the fats and oils are not particularly limited, and examples thereof include plant-derived fats and oils such as soybean oil, sesame oil, olive oil, coconut oil, rice oil, cottonseed oil, sunflower oil, corn oil, benflower oil, and rapeseed oil. .
- the manufactured fats and oils can be widely used for household use and industrial use, and can also be used as a raw material for biodiesel fuel. That is, according to the present invention, a plant body into which the protein phosphatase 2C gene and the glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene are introduced or the expression control region of the endogenous gene is modified is used.
- the above-described fats and oils for home use or industrial use, biodiesel fuel, and the like can be produced at low cost.
- the protein phosphatase 2C gene used in the present invention is presumed to be involved in information transmission related to gibberellic acid and abscisic acid among plant hormones, and is considered to be less related to these information transmission. An attempt was made to combine related glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase genes.
- the protein phosphatase 2C gene showed an additive effect in increasing the biomass amount and / or seed amount of the plant body into which both genes were introduced or the expression control region of the endogenous gene was modified.
- genes related to signal transduction related to gibberellic acid and abscisic acid and genes whose change in information transmission is not a major factor in increasing production of biomass and / or seed Can be expected. Examples of such genes include AINTEGUMENTA (Proc. Natl. Acad. Sci.
- T-DNA insertion site into the genome of the selected salt-tolerant Arabidopsis thaliana strain was determined by the TAIL-PCR method. First, young leaves were collected from cultivated Arabidopsis thaliana and ground under liquid nitrogen freezing. DNA was prepared using a DNA preparation kit (DNeasy Plant Mini Kit) manufactured by QIAGEN according to the standard protocol attached to the kit.
- Three kinds of specific primers TL1, TL2 and TL3 are set in the vicinity.
- TAIL-PCR (supervised by Isao Shimamoto, Takuji Sasaki, new edition, plant PCR) using the optional primer P1 under the following PCR reaction solution and reaction conditions Experimental protocol, 2000, 83-89pp, Shujunsha, Tokyo; Liu, YG and Whttier, RF, 1995, Genomics 25, 674-681; Liu, YG et al., Plant J., 8, 457-463, 1995 )
- primers TL1, TL2, TL3 and P1 are as follows.
- TL1 5'-TGC TTT CGC CAT TAA ATA GCG ACG G-3 '(SEQ ID NO: 24)
- TL2 5'-CGC TGC GGA CAT CTA CAT TTT TG-3 '(SEQ ID NO: 25)
- TL3 5'-TCC CGG ACA TGA AGC CAT TTA C-3 '(SEQ ID NO: 26)
- P1 5'-NGT CGA SWG ANA WGA A-3 '(SEQ ID NO: 27)
- n represents a, g, c or t (location: 1 and 11)
- s represents g or c (location: 7)
- w represents a or t is represented (location: 8 and 13).
- the first PCR reaction solution composition and reaction conditions are shown in Table 1 and Table 2, respectively.
- Tables 5 and 6 show the composition and reaction conditions of the third PCR reaction solution, respectively.
- the insertion site was the gene of Arabidopsis thaliana chromosome 3 [AGI (The Arabidopsis Genome Initiative gene code) Code: At3g05630].
- 5640PF1 (SEQ ID NO: 29): 5'-ACG CGT CGA CAT GGG ACA TTT CTC TTC CAT GTT CAA CGG-3 ' 5640PR1 (SEQ ID NO: 30): 5'-TGT ACA TGT ACA CTA TAG AGA TGG CGA CGA TGA AGA ATG G-3 ' 1-2-2.
- Template DNA was prepared from wild-type Arabidopsis thaliana, Col-0 ecotype, according to the method described. Phusion High-Fidelity DNA Polymerase (NEW ENGLAND BioLabs: manufactured by NEB) was used as an enzyme, and 5640PF1 and 5640PR1 were used as primers.
- the PCR reaction solution composition and reaction conditions are shown in Tables 7 and 8, respectively.
- the PCR amplification product was electrophoresed on a 2% agarose gel (TAE buffer), and the fragment was stained with ethidium bromide.
- TAE buffer 2% agarose gel
- the gel containing the target fragment was excised with a scalpel, and the target DNA fragment was eluted and purified using GFX PCR DNA and GEL Purification Kit (Amersham).
- Adenine was added to the obtained DNA fragment using A-Addition Kit (manufactured by QIAGEN).
- Amplified DNA added with adenine was ligated to the pCR2.1 vector for TA-Cloning using TOPO TA Cloning Kit (Invitrogen) and transformed into competent cells (E. coli TOP 10) attached to the kit. .
- the cells were cultured in LB medium supplemented with 50 ⁇ l / ml kanamycin, and transformants were selected.
- the emerged colonies were liquid-cultured in LB medium supplemented with 50 ⁇ l / ml kanamycin, and plasmid DNA was prepared from the obtained cells using Plasmid Mini Mini Kit manufactured by QIAGEN®.
- the resulting cloned vector containing the ORF fragment of PP2C (protein phosphatase 2C) gene (At3g05640) was sequenced and sequenced.
- Plant Expression Vector A construct was prepared by inserting a fragment containing the ORF of PP2C (protein phosphatase 2C) gene (At3g05640) into the plant expression vector pBI121 containing the omega sequence derived from tobacco mosaic virus.
- PP2C protein phosphatase 2C
- the pCR2.1 vector obtained by cloning the fragment containing the ORF of the PP2C (protein phosphatase 2C) gene (At3g05640) was treated with restriction enzymes SalI and BsrGI.
- pBI121 containing the omega sequence was similarly treated with restriction enzymes SalI and BsrGI. These restriction enzyme digests were subjected to 0.8% agarose gel electrophoresis, and fragments containing about 2700 bp PP2C (protein phosphatase 2C) ORF (At3g05640) from the gel using GFXGFPCR DNA and GEL Band Purification Kit (Amersham) And pBI121 containing omega sequence were separated and purified, respectively.
- the vector: insert ratio was mixed to 1:10, and equal amounts of TaKaRa Ligation A ligation reaction was performed overnight at 16 ° C. using kit ver.2 (manufactured by Takara Bio Inc.).
- the total amount of the reaction solution was added to 100 ⁇ l of competent cells (E. coli strain DH5 ⁇ , manufactured by TOYOBO), and transformation was performed according to the protocol attached to the kit. Apply to LB agar medium containing 50 ⁇ g / ml kanamycin and culture overnight. Appearance colonies are liquid-cultured in LB medium supplemented with 50 ⁇ g / ml kanamycin. From the obtained cells, Plasmid Mini Kit (manufactured by QIAGEN) ) was used to prepare plasmid DNA.
- Plasmid Mini Kit manufactured by QIAGEN
- the obtained expression vector obtained by subcloning the fragment containing the ORF of the PP2C (protein phosphatase 2C) gene (At3g05640) was subjected to nucleotide sequencing and sequence analysis.
- Transformants were selected on a kanamycin-containing medium, and T1 generation plants were produced by self-pollination to obtain T2 seeds.
- FIG. 5 shows a photograph of the above-ground part of a transformed plant into which a wild-type and PP2C (protein phosphatase 2C) gene (At3g05640) ORF-containing fragment was introduced. Moreover, as a result, the result of having measured the total biomass amount and seed amount of the above-ground part is shown in FIG.6 and FIG.7.
- PP2C protein phosphatase 2C
- Example 1 a glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene (hereinafter referred to as FBA1 gene) was further introduced into a transformed plant into which a PP2C (protein phosphatase 2C) gene (At3g05640) was introduced. A transformed plant was produced.
- FBA1 gene glutathione-binding plastid-type fructose 1,6-bisphosphate aldolase gene
- Plants are vermiculite (Asahi Kogyo Co., Ltd.), Kureha culture soil (Kureha Horticultural Culture, Kureha Chemical Co., Ltd.) and vermiculite from a bottom in a square plastic pot (6.5 x 6.5 x 5 cm): The seeds were sown on soil in 3 layers at a ratio of 2: 1 and grown under conditions of a growth temperature of 22 ° C. and long days (16-h light / 8-h dark).
- the full-length cDNA was amplified as two fragments by PCR, and each fragment was amplified by pGEM-T vector ( TA-cloning into Promega).
- the fragment containing the FBA1 gene and NOS terminator incorporated into the pBI121 vector was excised with XbaI and EcoRI and incorporated into a pBluscriptII (SK +) vector (Stratagene) treated with XbaI and EcoRI. Thereafter, a fragment containing the FBA1 gene and NOS terminator was excised with XbaI and KpnI and incorporated into a pMAT137-Hm vector treated with XbaI and KpnI.
- Transformants were selected on a hygromycin-containing medium, T1 generation plants were produced by self-pollination, and T2 seeds were obtained.
- the above-ground plant body was put in a paper bag and dried for 2 weeks under conditions of 22 ° C. and 60% humidity, and then the total biomass was weighed with an electronic balance.
- FIG. 8 shows a photograph of the above-ground part of a plant in which the FBA1 gene (At2g01140) has been introduced into the plant.
- the biomass amount shown in FIG. 9 has measured the biomass amount for every pot containing three individuals about six pots, and has shown the average value.
- the size of the above-ground part is wild type and the transformed plant body into which the FBA1 gene (At2g01140) is introduced into the transformed plant body into which the ORF-containing fragment of PP2C (protein phosphatase 2C) gene (At3g05640) is introduced. It was clarified that it was improved compared to the transformed plant into which the ORF-containing fragment of PP2C (protein phosphatase 2C) gene (At3g05640) was introduced.
- the total biomass amount in the above-ground part is PP2C in the transformed plant into which the FBA1 gene (At2g01140) is introduced into the transformed plant into which the fragment containing the ORF of the PP2C (protein phosphataset2C) gene (At3g05640) is introduced. It was revealed that this was improved by about 8 to 14% compared to the transformed plant into which the ORF-containing fragment of the (protein phosphatase 2C) gene (At3g05640) was introduced.
Abstract
Description
(b)配列番号5に示すアミノ酸配列において1又は複数個のアミノ酸が欠失、置換、付加又は挿入されたアミノ酸配列を含み、プロテインホスファターゼ2C活性を有するタンパク質
(c)配列番号4に示す塩基配列の相補的な塩基配列からなるポリヌクレオチドに対してストリンジェントな条件下においてハイブリダイズするポリヌクレオチドによってコードされプロテインホスファターゼ2C活性を有するタンパク質
また、本発明において、上記機能的に等価な遺伝子は、シロイヌナズナ以外の生物由来のプロテインホスファターゼ2C遺伝子を挙げることができる。シロイヌナズナ以外の生物としては、イネ(Oryza sativa)、ブラック・コットンウッド(Populus trichocarpa)、ヨーロッパブドウ(Vitis vinifera)、タルウマゴヤシ(Medicago truncatula)、アルファルファ(Medicago sativa)、ヒメツリカネゴケ(Physcomitrella patens)、アイスプラント(Mesembryanthemum crystallinum)、コナミドリムシ(Chlamydomonas reinhardtii)、トウモロコシ(Zea mays)、アブラナ(Brassica rapa)、トマト(Solanum lycopersicum)、ミゾホオズキ(Mimulus guttatus)、単細胞紅藻類のCyanidioschyzon merolaeからなる群から選ばれる一種を挙げることができる。
(b)配列番号32に示すアミノ酸配列において1又は複数個のアミノ酸が欠失、置換、付加又は挿入されたアミノ酸配列を含み、グルタチオンが結合することによりフルクトース1,6-ビスリン酸アルドラーゼ活性を示すタンパク質
(c)配列番号31に示す塩基配列の相補的な塩基配列からなるポリヌクレオチドに対してストリンジェントな条件下においてハイブリダイズするポリヌクレオチドによってコードされ、グルタチオンが結合することによりフルクトース1,6-ビスリン酸アルドラーゼ活性を示すタンパク質
また、本発明において、上記機能的に等価な遺伝子は、シロイヌナズナ以外の生物由来のグルタチオン結合性プラスチド型フルクトース1,6-ビスリン酸アルドラーゼ遺伝子を挙げることができる。シロイヌナズナ以外の生物は、イネ(Oryza sativa)、ヨーロッパブドウ(Vitis vinifera)、トウゴマ(Ricinus communis)、ブラック・コットンウッド(Populus trichocarpa)、シトカスプルース(Picea sitchensis)及びトウモロコシ(Zea mays)からなる群から選ばれる一種を挙げることができる。
本発明において、プロテインホスファターゼ2C遺伝子は、配列番号1~3に示すアミノ酸配列からなる3つの共通配列をN末端側からこの順で有するプロテインホスファターゼ2Cをコードする。なお、参考文献(TRENDS in Plant Science Vol.9 No.5 May 2004 Pages 236-243)の第237頁に挙げられたFigure1.topographic cladogramにおいてグループEとして分類された遺伝子群は、配列番号1~3に示すアミノ酸配列からなる3つの共通配列をN末端側からこの順で有するプロテインホスファターゼ2Cをコードする遺伝子である。なお、参考文献によれば、シロイヌナズナにおいては、76個のプロテインホスファターゼ2C遺伝子が予測されており、これら遺伝子についてT-Coffeeソフトウェア(参考文献;Notredame, C. et al. 2000 T-Coffee: a novel method for fast and accurate multiple sequence alignment. J. Mol. Biol. 302, 205-247)を用いて系統樹を作成した結果が参考文献のFigure1として開示されている。この系統樹において、グループEに分類されたプロテインホスファターゼ2C遺伝子は、配列番号1~3に示すアミノ酸配列からなる3つの共通配列をN末端側からこの順で有するプロテインホスファターゼ2Cをコードしている。配列番号1~3に示すアミノ酸配列からなる3つの共通配列は、上述した分類におけるグループEに特徴的な配列であって、他のグループとの明確な区別基準となる配列である。
このグループは、上記参考文献(1)で示された中性非極性アミノ酸のうち、脂肪属性の疎水性側鎖をもつアミノ酸のグループであり、V(Val、バリン)、L(Leu、ロイシン)、I(Ile、イソロイシン)及びM(Met、メチオニン)から構成される。参考文献(1)による中性非極性アミノ酸と分類されるもののうちFGACWPは以下理由で、この「脂肪族疎水性アミノ酸グループ」には含めない。G(Gly、グリシン)やA(Ala、アラニン)はメチル基以下の大きさで非極性の効果が弱いからである。C(Cys、システイン)はS-S結合に重要な役目を担う場合があり、また、酸素原子や窒素原子と水素結合を形成する特性があるからである。F(Phe、フェニルアラニン)やW(Trp、トリプトファン)は側鎖がとりわけ大きな分子量をもち、かつ、芳香族の効果が強いからである。P(Pro、プロリン)はイミノ酸効果が強く、ポリペプチドの主鎖の角度を固定してしまうからである。
このグループは、中性極性アミノ酸のうちヒドロキシメチレン基を側鎖に持つアミノ酸のグループであり、S(Ser、セリン)とT(Thr、スレオニン)から構成される。SとTの側鎖に存在する水酸基は、糖の結合部位であるため、あるポリペプチド(タンパク質)が特定の活性を持つために重要な部位である場合が多い。
このグループは、酸性であるカルボキシル基を側鎖に持つアミノ酸のグループであり、D(Asp、アスパラギン酸)とE(Glu、グルタミン酸)から構成される。
このグループは、塩基性アミノ酸のグループであり、K(Lys、リジン)とR(Arg、アルギニン)から構成される。これらKとRは、pHの広い範囲で正に帯電し塩基性の性質をもつ。一方、塩基性アミノ酸に分類されるH(His、ヒスチジン)はpH7においてほとんどイオン化されないので、このグループには分類されない。
このグループは、全てα位の炭素元素に側鎖としてメチレン基が結合しその先に極性基を有すると言う特徴を持つ。非極性基であるメチレン基の物理的大きさが酷似している特徴を持ち、N(Asn、アスパラギン、極性基はアミド基)、D(Asp、アスパラギン酸、極性基はカルボキシル基)とH(His、ヒスチジン、極性基はイミダゾール基)から成る。
6)ジメチレン基=極性基(EKQRグループ)
このグループは、全てα位の炭素元素に側鎖としてジメチレン基以上の直鎖炭化水素が結合しその先に極性基を有すると言う特徴を持つ。非極性基であるジメチレン基の物理的大きさが酷似している特徴を持つ。E(Glu、グルタミン酸、極性基はカルボキシル基)、K(Lys、リジン、極性基はアミノ基)、Q(Gln、グルタミン、極性基はアミド基)、R(Arg、アルギニン、極性基はイミノ基とアミノ基)から成る。
このグループには、側鎖にベンゼン核を持つ芳香族アミノ酸であり、芳香族特有の化学的性質を特徴とする。F(Phe、フェニルアラニン)、Y(Tyr、チロシン)、W(Trp、トリプトファン)から成る。
このグループには、側鎖に環状構造を持つと同時に極性も持つアミノ酸で、H(H、ヒスチジン、環状構造と極性基は共にイミダゾール基)、Y(Tyr、チロシン、環状構造はベンゼン核で極性基は水酸基)から成る。
本発明において、グルタチオン結合性プラスチド型フルクトース1,6-ビスリン酸アルドラーゼ遺伝子は、グルタチオンに結合することで活性が制御され、葉緑体等のプラスチド内においてフルクトース1,6-ビスリン酸をジヒドロキシアセトンリン酸とグリセルアルデヒド3-リン酸とに変換する反応(可逆反応)を触媒する活性を有する酵素をコードする遺伝子である。グルタチオン結合性プラスチド型フルクトース1,6-ビスリン酸アルドラーゼ遺伝子は、上記活性を有する酵素をコードする遺伝子であれば、如何なる植物由来の遺伝子であっても良いし、植物から単離した遺伝子に突然変異を導入した変異型遺伝子であっても良い。
発現ベクターは、植物体内で発現を可能とするプロモーターと、上述したプロテインホスファターゼ2C遺伝子及びグルタチオン結合性プラスチド型フルクトース1,6-ビスリン酸アルドラーゼ遺伝子を含むように構築する。なお、プロテインホスファターゼ2C遺伝子を含む発現ベクターと、グルタチオン結合性プラスチド型フルクトース1,6-ビスリン酸アルドラーゼ遺伝子を含む発現ベクターとを別個に準備しても良い。
上述した発現ベクターは、一般的な形質転換方法によって対象の植物内に導入される。発現ベクターを植物細胞に導入する方法(形質転換方法)は特に限定されるものではなく、植物細胞に応じた適切な従来公知の方法を用いることができる。具体的には、例えば、アグロバクテリウムを用いる方法や直接植物細胞に導入する方法を用いることができる。アグロバクテリウムを用いる方法としては、例えば、Bechtold, E., Ellis, J. and Pelletier, G. (1993) In Planta Agrobacterium-mediated gene transfer by infiltration of adult Arabidopsis plants. C.R. Acad. Sci. Paris Sci. Vie, 316, 1194-1199. あるいは、Zyprian E, Kado Cl, Agrobacterium-mediated plant transformation by novel mini-T vectors in conjunction with a high-copy vir region helper plasmid. Plant Molecular Biology, 1990, 15(2), 245-256.に記載された方法を用いることができる。
上述した形質転換処理後、植物体のなかから適切な形質転換体を選抜する選抜工程を、従来公知の方法で行うことができる。選抜の方法は特に限定されるものではなく、例えば、ハイグロマイシン耐性等の薬剤耐性を基準として選抜してもよいし、形質転換体を育成した後に、植物体そのもの、または任意の器官や組織の重量を測定して野生型と比較して有意に増産しているものを選抜してもよい。
1.材料および方法
1-1.実験材料
実験材料にはシロイヌナズナ変異体(Activation-tag lines: Weigel T-DNA lines、20072系統)の種子を用いた。なお、種子はNottingham Arabidopsis Stock Centre(NASC) より購入した。実験材料として使用した種子についてはWeigel, D. et al., 2000, Plant Physiol. 122, 1003-1013を参考とすることができる。
1-2-1.塩耐性変異体の選抜
Weigel T-DNA linesの種子を、NaCl 125 mMあるいは150 mMを含む改変MS寒天(1%)培地〔B5培地のビタミン、ショ糖10 g/l、寒天(細菌培地用;和光純薬工業社製)8 g/L〕に無菌播種し、22℃、30~100μmol/m2/secの照明下(16時間明期/8時間暗期のサイクル)で培養した。播種後2~4週間後、塩耐性変異体候補を選抜した。なお、MS培地はMurashige, T. et al., 1962, Physiol. Plant. 15, 473-497を参照する。また、B5培地についてはGamborg, O.L. et al. , 1968, Experimental Cell Research 50, 151-158を参照する。
選抜した耐塩性シロイヌナズナ系統のゲノムへのT-DNA挿入部位を、TAIL-PCR法により決定した。まず、栽培したシロイヌナズナから幼葉を採取し、液体窒素凍結下で粉砕した。QIAGEN社製DNA調製キット( DNeasy Plant Mini Kit)を用いてキット添付の標準プロトコルに従ってDNAを調製した。
Weigel T-DNA linesで用いられているアクティベーションタギング用ベクター(pSKI015 : GenBank accession No.AF187951)の左のT-DNA配列(T-DNA left border)付近に3種類の特異的プライマーTL1、TL2及びTL3を設定し、任意プライマーP1を用いて、以下のPCR反応液及び反応条件下でTAIL-PCR(島本功、佐々木卓治監修, 新版, 植物のPCR実験プロトコール, 2000, 83-89pp, 秀潤社, 東京 ; Liu, Y.G. and Whttier, R.F., 1995, Genomics 25, 674-681 ; Liu, Y.G. et al., Plant J., 8, 457-463, 1995)を行い、T-DNAに隣接するゲノムDNAを増幅した。
TL2: 5'-CGC TGC GGA CAT CTA CAT TTT TG-3'(配列番号25)
TL3: 5'-TCC CGG ACA TGA AGC CAT TTA C-3'(配列番号26)
P1: 5'-NGT CGA SWG ANA WGA A-3'(配列番号27)
なお、配列番号25において、nは、a、g、c又はtを表し(存在位置:1及び11)、またsは、g又はcを表し(存在位置:7)、さらにwは、a又はtを表す(存在位置:8及び13)。
アクティベートされている遺伝子を、1-2-3.により判明したT-DNA挿入部位(At3g05630)近傍10Kb以内に存在する推定Open reading frame(ORF)遺伝子の配列から予測した。
1-2-4.でアクティベート(活性化)されていると予測したPP2C(protein phosphatase 2C)遺伝子(At3g05640)のORF領域を含む断片を増幅するために、TAIR(http://www.arabidopsis.org/home.html)で公開されている配列情報を基にPCR用プライマー5640PF1及び5640PR1を設計・合成した。なおこれらプライマーの末端には、発現ベクターへ導入するときに必要となる制限酵素サイト(Bsr G IまたはSal I)を付加するように設計した。
5'-ACG CGT CGA CAT GGG ACA TTT CTC TTC CAT GTT CAA CGG-3'
5640PR1(配列番号30):
5'-TGT ACA TGT ACA CTA TAG AGA TGG CGA CGA CGA TGA AGA ATG G-3'
1-2-2.記載の方法に従って、野生型シロイヌナズナ、Col-0エコタイプから鋳型DNAを調製した。酵素としてPhusion High-Fidelity DNA Polymerase(NEW ENGLAND BioLabs:NEB社製)、プライマーとして、上記5640PF1および5640PR1を用いた。PCRの反応液組成、および反応条件をそれぞれ表7及び8に示す。
タバコモザイクウイルス由来のomega配列を含む植物発現用ベクターpBI121に、PP2C(protein phosphatase 2C)遺伝子(At3g05640)のORFを含む断片を挿入したコンストラクトの作製を行った。
1-2-6.で作製した植物発現用ベクターをエレクトロポレーション法(Plant Molecular Biology Mannal, Second Edition , B. G. Stanton and A. S. Robbert, Kluwer Acdemic Publishers 1994)により、アグロバクテリウム・ツメファシエンス(Agrobacterium tumefaciens)C58C1株に導入した。次いで植物発現用ベクターが導入されたアグロバクテリウム・ツメファシエンスを、Cloughらにより記載された浸潤法(Steven J. Clough and Andrew F. Bent, 1998, The Plant Journal 16, 735-743)により、野生型シロイヌナズナ エコタイプCol-0に導入した。
1-2-7.で作製したT2種子を無菌播種し、バーミキュライト混合土を入れた直径50mmのポットに移植した。比較対象として非組換えシロイヌナズナを移植した。これを22℃、16時間明期8時間暗期、光強度約30~45μmol m-2 s-1で栽培し移植後合計11週間栽培した。栽培後、地上部の植物体を紙袋に入れ、22℃、湿度60%の条件で2週間乾燥させた後に、全バイオマス量及び種子量を電子天秤で秤量した。
上記1-2-8.の結果として、野生型及びPP2C(protein phosphatase 2C)遺伝子(At3g05640)のORFを含む断片を導入した形質転換植物体の地上部を撮影した写真を図5に示す。また、結果として、地上部の全バイオマス量及び種子量を測定した結果を図6及び図7に示す。
本実施例1では、PP2C(protein phosphatase 2C)遺伝子(At3g05640)を導入した形質転換植物に対して、更にグルタチオン結合性プラスチド型フルクトース1,6-ビスリン酸アルドラーゼ遺伝子(以下、FBA1遺伝子)を導入した形質転換植物を作製した。
2-1.実験材料
実験材料には、1-2-7.で作製したPP2C(protein phosphatase 2C)遺伝子(At3g05640)のORFを含む断片を導入した形質転換植物体のT3世代以降の種子を用いた。野生型シロイヌナズナは、エコタイプCol-0を用いた。
2-2-1.FBA1遺伝子(At2g01140)の取得
4週齢のシロイヌナズナの野生型Columbia(Col-0)からトータルRNAを単離し、Prost arfirststrand RT-PCRキット(Stratagene社製)を用いてRT-PCR(テンプレートRNA量5.0μg)を行い、cDNAを作製した。
1R-1:5’-ATCTGCAACGGTCTCGGGAGA-3’(配列番号45)
1F-2:5’-GTGTGGTCCGAGGTGTTCTTCT-3’(配列番号46)
1R-2:5’-GAGCTCGAGTAGGTGTAACCCTTG-3’(配列番号47)
2つの断片をBstpIサイトで融合し、完全長cDNAを含むベクター(pGEM-FBA1)を構築した。形質転換植物を作出するためにpGEM-FBAlを制限酵素BamHIとSacIで処理後、断片をpBI121ベクターに導入した。
植物発現用ベクターpMAT137-HM(Matsuoka K. and Nakamura K., 1991, Proc. Natl. Acad. Sci. U S A 88, 834-838)に、2-2-1.で取得したFBA1遺伝子(At2g01140)を含む断片を挿入したコンストラクトの作製を行った。
2-2-2.で作製した植物発現用ベクターpMAT137-Hmをエレクトロポレーション法(Plant Molecular Biology Mannal, Second Edition , B. G. Stanton and A. S. Robbert, Kluwer Acdemic Publishers 1994)により、アグロバクテリウム・ツメファシエンス(Agrobacterium tumefaciens)C58C1株に導入した。次いで、植物発現用ベクターが導入されたアグロバクテリウム・ツメファシエンスを、Cloughらにより記載された浸潤法(Steven J. Clough and Andrew F. Bent, 1998, The Plant Journal 16, 735-743)により、野生型シロイヌナズナエコタイプCol-0に導入した。
2-2-3.で作製したT2種子を播種し、正方形のプラスチックポット(6.5×6.5×5cm)の中に底からバーミキュライト(旭工業社製)、クレハ培養土(クレハ園芸培土、呉羽化学社製)、バーミキュライトを2:2:1の割合で3層に入れた土壌に播種し、生育温度22℃、長日(16-h明/8-h暗)条件で生育させた。対象には参考例1における1-2-7.で作製したPP2C遺伝子を導入した形質転換植物体を用いた。これを22℃、16時間明期8時間暗期、光強度約70~100μmol m-2 s-1で栽培し移植後合計11週間栽培した。栽培後、地上部の植物体を紙袋に入れ、22℃、湿度60%の条件で2週間乾燥させた後に、全バイオマス量を電子天秤で秤量した。
上記2-2-4.の結果として、野生型、PP2C(protein phosphatase 2C)遺伝子(At3g05640)のORFを含む断片を導入した形質転換植物体及びPP2C(protein phosphatase 2C)遺伝子(At3g05640)のORFを含む断片を導入した形質転換植物体にFBA1遺伝子(At2g01140)を導入した植物体の地上部を撮影した写真を図8に示す。また、結果として、地上部の全バイオマス量を測定した結果を図9に示す。なお、図9に示したバイオマス量は、3個体を含むポット毎のバイオマス量を6個のポットについて測定し、その平均値を示している。
Claims (51)
- 配列番号1~3に示すアミノ酸配列からなる3つの共通配列をN末端側からこの順で有するプロテインホスファターゼ2Cをコードする遺伝子及びグルタチオン結合性プラスチド型フルクトース1,6-ビスリン酸アルドラーゼをコードする遺伝子を導入した、又は内在する当該遺伝子の発現制御領域を改変した植物体。
- 上記プロテインホスファターゼ2Cをコードする遺伝子は、At1g03590-AtPP2C6-6、At1g16220、At1g79630、At5g01700、At3g02750、At5g36250、At5g26010、At4g32950、At3g16800、At3g05640、At5g27930-AtPP2C6-7、At2g20050及びAt3g06270からなる群から選ばれる少なくとも1種の遺伝子若しくは当該遺伝子に機能的に等価な遺伝子であることを特徴とする請求項1記載の植物体。
- 上記プロテインホスファターゼ2Cをコードする遺伝子が、以下の(a)~(c)のいずれかのタンパク質をコードすることを特徴とする請求項1記載の植物体。
(a)配列番号5に示すアミノ酸配列を含むタンパク質
(b)配列番号5に示すアミノ酸配列において1又は複数個のアミノ酸が欠失、置換、付加又は挿入されたアミノ酸配列を含み、プロテインホスファターゼ2C活性を有するタンパク質
(c)配列番号4に示す塩基配列の相補的な塩基配列からなるポリヌクレオチドに対してストリンジェントな条件下においてハイブリダイズするポリヌクレオチドによってコードされプロテインホスファターゼ2C活性を有するタンパク質 - 上記機能的に等価な遺伝子は、シロイヌナズナ以外の生物由来のプロテインホスファターゼ2C遺伝子であることを特徴とする請求項2記載の植物体。
- 上記シロイヌナズナ以外の生物は、イネ(Oryza sativa)、ブラック・コットンウッド(Populus trichocarpa)、ヨーロッパブドウ(Vitis vinifera)、タルウマゴヤシ(Medicago truncatula)、アルファルファ(Medicago sativa)、ヒメツリカネゴケ(Physcomitrella patens)、アイスプラント(Mesembryanthemum crystallinum)、コナミドリムシ(Chlamydomonas reinhardtii)、トウモロコシ(Zea mays)、アブラナ(Brassica rapa)、トマト(Solanum lycopersicum)、ミゾホオズキ(Mimulus guttatus)、単細胞紅藻類のCyanidioschyzon merolaeからなる群から選ばれる一種であることを特徴とする請求項4記載の植物体。
- 上記グルタチオン結合性プラスチド型フルクトース1,6-ビスリン酸アルドラーゼをコードする遺伝子は、At2g01140遺伝子若しくは当該遺伝子に機能的に等価な遺伝子であることを特徴とする請求項1記載の植物体。
- 上記グルタチオン結合性プラスチド型フルクトース1,6-ビスリン酸アルドラーゼをコードする遺伝子が、以下の(a)~(c)のいずれかのタンパク質をコードすることを特徴とする請求項1記載の植物体。
(a)配列番号32に示すアミノ酸配列を含むタンパク質
(b)配列番号32に示すアミノ酸配列において1又は複数個のアミノ酸が欠失、置換、付加又は挿入されたアミノ酸配列を含み、グルタチオンが結合することによりフルクトース1,6-ビスリン酸アルドラーゼ活性を示すタンパク質
(c)配列番号31に示す塩基配列の相補的な塩基配列からなるポリヌクレオチドに対してストリンジェントな条件下においてハイブリダイズするポリヌクレオチドによってコードされ、グルタチオンが結合することによりフルクトース1,6-ビスリン酸アルドラーゼ活性を示すタンパク質 - 上記機能的に等価な遺伝子は、シロイヌナズナ以外の生物由来のグルタチオン結合性プラスチド型フルクトース1,6-ビスリン酸アルドラーゼ遺伝子であることを特徴とする請求項6記載の植物体。
- 上記シロイヌナズナ以外の生物は、イネ(Oryza sativa)、ヨーロッパブドウ(Vitis vinifera)、トウゴマ(Ricinus communis)、ブラック・コットンウッド(Populus trichocarpa)、シトカスプルース(Picea sitchensis)及びトウモロコシ(Zea mays)からなる群から選ばれる一種であることを特徴とする請求項8記載の植物体。
- 双子葉植物であることを特徴とする請求項1乃至9いずれか一項記載の植物体。
- アブラナ科植物であることを特徴とする請求項1乃至9いずれか一項記載の植物体。
- シロイヌナズナであることを特徴とする請求項1乃至9いずれか一項記載の植物体。
- アブラナであることを特徴とする請求項1乃至9いずれか一項記載の植物体。
- 単子葉植物であることを特徴とする請求項1乃至9いずれか一項記載の植物体。
- イネ科植物であることを特徴とする請求項1乃至9いずれか一項記載の植物体。
- イネであることを特徴とする請求項1乃至9いずれか一項記載の植物体。
- サトウキビであることを特徴とする請求項1乃至9いずれか一項記載の植物体。
- 配列番号1~3に示すアミノ酸配列からなる3つの共通配列をN末端側からこの順で有するプロテインホスファターゼ2Cをコードする遺伝子及びグルタチオン結合性プラスチド型フルクトース1,6-ビスリン酸アルドラーゼをコードする遺伝子を導入する、又は内在する当該遺伝子の発現制御領域を改変する、バイオマス量及び/又は種子量を増産させる方法。
- 上記プロテインホスファターゼ2Cをコードする遺伝子は、At1g03590-AtPP2C6-6、At1g16220、At1g79630、At5g01700、At3g02750、At5g36250、At5g26010、At4g32950、At3g16800、At3g05640、At5g27930-AtPP2C6-7、At2g20050及びAt3g06270からなる群から選ばれる少なくとも1種の遺伝子若しくは当該遺伝子に機能的に等価な遺伝子であることを特徴とする請求項18記載の方法。
- 上記プロテインホスファターゼ2Cをコードする遺伝子が、以下の(a)~(c)のいずれかのタンパク質をコードすることを特徴とする請求項18記載の方法。
(a)配列番号5に示すアミノ酸配列を含むタンパク質
(b)配列番号5に示すアミノ酸配列において1又は複数個のアミノ酸が欠失、置換、付加又は挿入されたアミノ酸配列を含み、プロテインホスファターゼ2C活性を有するタンパク質
(c)配列番号4に示す塩基配列の相補的な塩基配列からなるポリヌクレオチドに対してストリンジェントな条件下においてハイブリダイズするポリヌクレオチドによってコードされプロテインホスファターゼ2C活性を有するタンパク質 - 上記機能的に等価な遺伝子は、シロイヌナズナ以外の生物由来のプロテインホスファターゼ2C遺伝子であることを特徴とする請求項19記載の方法。
- 上記シロイヌナズナ以外の生物は、イネ(Oryza sativa)、ブラック・コットンウッド(Populus trichocarpa)、ヨーロッパブドウ(Vitis vinifera)、タルウマゴヤシ(Medicago truncatula)、アルファルファ(Medicago sativa)、ヒメツリカネゴケ(Physcomitrella patens)、アイスプラント(Mesembryanthemum crystallinum)、コナミドリムシ(Chlamydomonas reinhardtii)、トウモロコシ(Zea mays)、アブラナ(Brassica rapa)、トマト(Solanum lycopersicum)、ミゾホオズキ(Mimulus guttatus)、単細胞紅藻類のCyanidioschyzon merolaeからなる群から選ばれる一種であることを特徴とする請求項21記載の方法。
- 上記グルタチオン結合性プラスチド型フルクトース1,6-ビスリン酸アルドラーゼをコードする遺伝子は、At2g01140遺伝子若しくは当該遺伝子に機能的に等価な遺伝子であることを特徴とする請求項18記載の方法。
- 上記グルタチオン結合性プラスチド型フルクトース1,6-ビスリン酸アルドラーゼをコードする遺伝子が、以下の(a)~(c)のいずれかのタンパク質をコードすることを特徴とする請求項18記載の方法。
(a)配列番号32に示すアミノ酸配列を含むタンパク質
(b)配列番号32に示すアミノ酸配列において1又は複数個のアミノ酸が欠失、置換、付加又は挿入されたアミノ酸配列を含み、グルタチオンが結合することによりフルクトース1,6-ビスリン酸アルドラーゼ活性を示すタンパク質
(c)配列番号31に示す塩基配列の相補的な塩基配列からなるポリヌクレオチドに対してストリンジェントな条件下においてハイブリダイズするポリヌクレオチドによってコードされ、グルタチオンが結合することによりフルクトース1,6-ビスリン酸アルドラーゼ活性を示すタンパク質 - 上記機能的に等価な遺伝子は、シロイヌナズナ以外の生物由来のグルタチオン結合性プラスチド型フルクトース1,6-ビスリン酸アルドラーゼ遺伝子であることを特徴とする請求項23記載の方法。
- 上記シロイヌナズナ以外の生物は、イネ(Oryza sativa)、ヨーロッパブドウ(Vitis vinifera)、トウゴマ(Ricinus communis)、ブラック・コットンウッド(Populus trichocarpa)、シトカスプルース(Picea sitchensis)及びトウモロコシ(Zea mays)からなる群から選ばれる一種であることを特徴とする請求項25記載の方法。
- 双子葉植物であることを特徴とする請求項18乃至26いずれか一項記載の方法。
- アブラナ科植物であることを特徴とする請求項18乃至26いずれか一項記載の方法。
- シロイヌナズナであることを特徴とする請求項18乃至26いずれか一項記載の方法。
- アブラナであることを特徴とする請求項18乃至26いずれか一項記載の方法。
- 単子葉植物であることを特徴とする請求項18乃至26いずれか一項記載の方法。
- イネ科植物であることを特徴とする請求項18乃至26いずれか一項記載の方法。
- イネであることを特徴とする請求項18乃至26いずれか一項記載の方法。
- サトウキビであることを特徴とする請求項18乃至26いずれか一項記載の方法。
- 配列番号1~3に示すアミノ酸配列からなる3つの共通配列をN末端側からこの順で有するプロテインホスファターゼ2Cをコードする遺伝子及びグルタチオン結合性プラスチド型フルクトース1,6-ビスリン酸アルドラーゼをコードする遺伝子を導入した、又は内在する当該遺伝子の発現制御領域を改変した形質転換植物を準備する工程と、
前記形質転換植物の後代植物のバイオマス量及び/又は種子量を測定し、当該バイオマス量及び/又は種子量が有意に向上した系統を選抜する工程とを含む、植物体の製造方法。 - 上記プロテインホスファターゼ2Cをコードする遺伝子は、At1g03590-AtPP2C6-6、At1g16220、At1g79630、At5g01700、At3g02750、At5g36250、At5g26010、At4g32950、At3g16800、At3g05640、At5g27930-AtPP2C6-7、At2g20050及びAt3g06270からなる群から選ばれる少なくとも1種の遺伝子若しくは当該遺伝子に機能的に等価な遺伝子であることを特徴とする請求項35記載の製造方法。
- 上記プロテインホスファターゼ2Cをコードする遺伝子が、以下の(a)~(c)のいずれかのタンパク質をコードすることを特徴とする請求項35記載の製造方法。
(a)配列番号5に示すアミノ酸配列を含むタンパク質
(b)配列番号5に示すアミノ酸配列において1又は複数個のアミノ酸が欠失、置換、付加又は挿入されたアミノ酸配列を含み、プロテインホスファターゼ2C活性を有するタンパク質
(c)配列番号4に示す塩基配列の相補的な塩基配列からなるポリヌクレオチドに対してストリンジェントな条件下においてハイブリダイズするポリヌクレオチドによってコードされプロテインホスファターゼ2C活性を有するタンパク質 - 上記機能的に等価な遺伝子は、シロイヌナズナ以外の生物由来のプロテインホスファターゼ2C遺伝子であることを特徴とする請求項36記載の製造方法。
- 上記シロイヌナズナ以外の生物は、イネ(Oryza sativa)、ブラック・コットンウッド(Populus trichocarpa)、ヨーロッパブドウ(Vitis vinifera)、タルウマゴヤシ(Medicago truncatula)、アルファルファ(Medicago sativa)、ヒメツリカネゴケ(Physcomitrella patens)、アイスプラント(Mesembryanthemum crystallinum)、コナミドリムシ(Chlamydomonas reinhardtii)、トウモロコシ(Zea mays)、アブラナ(Brassica rapa)、トマト(Solanum lycopersicum)、ミゾホオズキ(Mimulus guttatus)、単細胞紅藻類のCyanidioschyzon merolaeからなる群から選ばれる一種であることを特徴とする請求項38記載の製造方法。
- 上記グルタチオン結合性プラスチド型フルクトース1,6-ビスリン酸アルドラーゼをコードする遺伝子は、At2g01140遺伝子若しくは当該遺伝子に機能的に等価な遺伝子であることを特徴とする請求項35記載の製造方法。
- 上記グルタチオン結合性プラスチド型フルクトース1,6-ビスリン酸アルドラーゼをコードする遺伝子が、以下の(a)~(c)のいずれかのタンパク質をコードすることを特徴とする請求項35記載の製造方法。
(a)配列番号32に示すアミノ酸配列を含むタンパク質
(b)配列番号32に示すアミノ酸配列において1又は複数個のアミノ酸が欠失、置換、付加又は挿入されたアミノ酸配列を含み、グルタチオンが結合することによりフルクトース1,6-ビスリン酸アルドラーゼ活性を示すタンパク質
(c)配列番号31に示す塩基配列の相補的な塩基配列からなるポリヌクレオチドに対してストリンジェントな条件下においてハイブリダイズするポリヌクレオチドによってコードされ、グルタチオンが結合することによりフルクトース1,6-ビスリン酸アルドラーゼ活性を示すタンパク質 - 上記機能的に等価な遺伝子は、シロイヌナズナ以外の生物由来のグルタチオン結合性プラスチド型フルクトース1,6-ビスリン酸アルドラーゼ遺伝子であることを特徴とする請求項40記載の製造方法。
- 上記シロイヌナズナ以外の生物は、イネ(Oryza sativa)、ヨーロッパブドウ(Vitis vinifera)、トウゴマ(Ricinus communis)、ブラック・コットンウッド(Populus trichocarpa)、シトカスプルース(Picea sitchensis)及びトウモロコシ(Zea mays)からなる群から選ばれる一種であることを特徴とする請求項42記載の製造方法。
- 双子葉植物であることを特徴とする請求項35乃至43いずれか一項記載の製造方法。
- アブラナ科植物であることを特徴とする請求項35乃至43いずれか一項記載の製造方法。
- シロイヌナズナであることを特徴とする請求項35乃至43いずれか一項記載の製造方法。
- アブラナであることを特徴とする請求項35乃至43いずれか一項記載の製造方法。
- 単子葉植物であることを特徴とする請求項35乃至43いずれか一項記載の製造方法。
- イネ科植物であることを特徴とする請求項35乃至43いずれか一項記載の製造方法。
- イネであることを特徴とする請求項35乃至43いずれか一項記載の製造方法。
- サトウキビであることを特徴とする請求項35乃至43いずれか一項記載の製造方法。
Priority Applications (4)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| AU2010222106A AU2010222106B2 (en) | 2009-03-12 | 2010-03-10 | Gene for increasing the production of plant biomass and/or seeds and method for use thereof |
| US13/256,190 US9546376B2 (en) | 2009-03-12 | 2010-03-10 | Gene for increasing the production of plant biomass and/or seeds and method for use thereof |
| BRPI1009784-8A BRPI1009784A2 (pt) | 2009-03-12 | 2010-03-10 | Gene para aumentar a produção de biomassa vegetal e/ou sementes e método para aplicação das mesmas. |
| CN2010800113921A CN102395675A (zh) | 2009-03-12 | 2010-03-10 | 使植物的生物量和/或种子量增产的基因及其利用方法 |
Applications Claiming Priority (2)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| JP2009060154A JP5604657B2 (ja) | 2009-03-12 | 2009-03-12 | 植物のバイオマス量及び/又は種子量を増産させる遺伝子及びその利用方法 |
| JP2009-060154 | 2009-03-12 |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| WO2010104092A1 true WO2010104092A1 (ja) | 2010-09-16 |
Family
ID=42728381
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| PCT/JP2010/053939 WO2010104092A1 (ja) | 2009-03-12 | 2010-03-10 | 植物のバイオマス量及び/又は種子量を増産させる遺伝子及びその利用方法 |
Country Status (6)
| Country | Link |
|---|---|
| US (1) | US9546376B2 (ja) |
| JP (1) | JP5604657B2 (ja) |
| CN (1) | CN102395675A (ja) |
| AU (1) | AU2010222106B2 (ja) |
| BR (1) | BRPI1009784A2 (ja) |
| WO (1) | WO2010104092A1 (ja) |
Cited By (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2012039159A1 (ja) * | 2010-09-22 | 2012-03-29 | 国立大学法人奈良先端科学技術大学院大学 | 塊茎生産能または匍匐枝形成能が野生株に比して向上している匍匐枝形成植物の作製方法、当該方法によって作製された匍匐枝形成植物 |
Families Citing this family (5)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| BRPI0909344A2 (pt) * | 2008-03-14 | 2015-08-04 | Toyota Motor Co Ltd | Gene para aumentar a produção de biomassa vegetal e/ou sementes e método para o seu uso |
| JP5604657B2 (ja) | 2009-03-12 | 2014-10-08 | トヨタ自動車株式会社 | 植物のバイオマス量及び/又は種子量を増産させる遺伝子及びその利用方法 |
| US9238819B2 (en) * | 2011-05-04 | 2016-01-19 | Versitech Limited | Method for speeding up plant growth and improving yield by altering expression levels of kinases and phosphatases |
| JP6397610B2 (ja) | 2013-09-10 | 2018-09-26 | 株式会社豊田中央研究所 | 師部組織増産に適した植物体及びその利用 |
| CN115028696B (zh) * | 2021-02-20 | 2023-05-02 | 中国农业大学 | 一种与果实品质相关的蛋白及其编码基因的应用 |
Citations (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2007091634A1 (ja) * | 2006-02-09 | 2007-08-16 | Japan Science And Technology Agency | 成長性および病虫害抵抗性が向上した植物、並びにその作出方法 |
| WO2008072602A1 (ja) * | 2006-12-11 | 2008-06-19 | Japan Science And Technology Agency | 植物生長調整剤及びその利用 |
Family Cites Families (22)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| DE69434715D1 (de) | 1993-10-06 | 2006-06-01 | Univ New York | Methode zur herstellung von transgenen pflanzen, die eine erhöhte stickstoffaufnahme zeigen |
| WO1998010082A1 (en) | 1994-09-01 | 1998-03-12 | University Of Florida Research Foundation, Inc. | Materials and methods for increasing corn seed weight |
| US6252139B1 (en) | 1996-07-18 | 2001-06-26 | The Salk Institute For Biological Studies | Method of increasing growth and yield in plants |
| JP2001505410A (ja) | 1996-09-05 | 2001-04-24 | ユニバーシティー オブ フロリダ リサーチ ファウンデーション インク. | トウモロコシの種子重量を増加させるための材料および方法 |
| EA003425B1 (ru) | 1997-03-26 | 2003-04-24 | Кембридж Юниверсити Текникал Сервисиз Лимитед | Химерный ген, обеспечивающий экспрессию в растениях циклина d-типа, и способ получения растений с измененными ростовыми свойствами при использовании указанного гена |
| US7598361B2 (en) * | 1997-11-24 | 2009-10-06 | Monsanto Technology Llc | Nucleic acid molecules and other molecules associated with the sucrose pathway |
| US20100293663A2 (en) | 1998-06-16 | 2010-11-18 | Thomas La Rosa | Nucleic Acid Molecules and Other Molecules Associated with Plants and Uses Thereof for Plant Improvement |
| US20020040490A1 (en) | 2000-01-27 | 2002-04-04 | Jorn Gorlach | Expressed sequences of arabidopsis thaliana |
| EP1313867A2 (en) | 2000-08-24 | 2003-05-28 | The Scripps Research Institute | Stress-regulated genes of plants, transgenic plants containing same, and methods of use |
| AU2001289843A1 (en) * | 2001-08-28 | 2002-02-13 | Bayer Cropscience Ag | Polypeptides for identifying herbicidally active compounds |
| JP2005185101A (ja) | 2002-05-30 | 2005-07-14 | National Institute Of Agrobiological Sciences | 植物の全長cDNAおよびその利用 |
| US20070136839A1 (en) | 2003-10-14 | 2007-06-14 | Ceres, Inc. | Promoter, promoter control elements, and combinations, and uses thereof |
| JP2005130770A (ja) | 2003-10-30 | 2005-05-26 | Ajinomoto Co Inc | 植物体あたり得られるデンプン量が増加したバレイショおよびその作出法 |
| US7790956B2 (en) | 2004-04-02 | 2010-09-07 | Cropdesign N.V. | Plants having improved growth characteristics and methods for making the same by modulating expression of a nucleic acid sequence encoding a NAP1-like protein |
| EP2584033A1 (en) * | 2005-01-12 | 2013-04-24 | Monsanto Technology LLC | Genes and uses for plant improvement |
| CA2644273A1 (en) * | 2006-04-05 | 2008-03-27 | Metanomics Gmbh | Process for the production of a fine chemical |
| US20080261913A1 (en) | 2006-12-28 | 2008-10-23 | Idenix Pharmaceuticals, Inc. | Compounds and pharmaceutical compositions for the treatment of liver disorders |
| CN101583713B (zh) | 2007-01-16 | 2013-07-10 | 独立行政法人科学技术振兴机构 | 种子收量提高的植物 |
| BRPI0909344A2 (pt) * | 2008-03-14 | 2015-08-04 | Toyota Motor Co Ltd | Gene para aumentar a produção de biomassa vegetal e/ou sementes e método para o seu uso |
| JP5604657B2 (ja) | 2009-03-12 | 2014-10-08 | トヨタ自動車株式会社 | 植物のバイオマス量及び/又は種子量を増産させる遺伝子及びその利用方法 |
| JP5519192B2 (ja) | 2009-06-04 | 2014-06-11 | トヨタ自動車株式会社 | 種子のタンパク質含量を増産させる遺伝子及びその利用方法 |
| JP5250807B2 (ja) | 2009-09-11 | 2013-07-31 | トヨタ自動車株式会社 | 植物のバイオマス量及び/又は種子量を増産させる方法、バイオマス量及び/又は種子量を増産できる植物の製造方法 |
-
2009
- 2009-03-12 JP JP2009060154A patent/JP5604657B2/ja not_active Expired - Fee Related
-
2010
- 2010-03-10 CN CN2010800113921A patent/CN102395675A/zh active Pending
- 2010-03-10 US US13/256,190 patent/US9546376B2/en active Active
- 2010-03-10 WO PCT/JP2010/053939 patent/WO2010104092A1/ja active Application Filing
- 2010-03-10 BR BRPI1009784-8A patent/BRPI1009784A2/pt not_active Application Discontinuation
- 2010-03-10 AU AU2010222106A patent/AU2010222106B2/en not_active Ceased
Patent Citations (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2007091634A1 (ja) * | 2006-02-09 | 2007-08-16 | Japan Science And Technology Agency | 成長性および病虫害抵抗性が向上した植物、並びにその作出方法 |
| WO2008072602A1 (ja) * | 2006-12-11 | 2008-06-19 | Japan Science And Technology Agency | 植物生長調整剤及びその利用 |
Non-Patent Citations (4)
| Title |
|---|
| "Annual Meeting of the Molecular Biology Society of Japan Koen Yoshishu, 20 November 2009 (20.11.2009)", 20 November 2009, article HIROKI SUGIMOTO ET AL.: "Arabidopsis thaliana PP2C (AtPP2CF1) no Kino Kaiseki" * |
| "Japanese Society for Plant Cell and Molecular Biology Taikai- Symposium Koen Yoshishu, 08 August 2007 (08.08. 2007)", 8 August 2007, article KENJI KOMATSU ET AL.: "Physcomitrella patens ni Okeru Protein Phosphatase 2C o Kaishita ABA Signal Dentatsu no Kaiseki", pages: 143 * |
| KEN'ICHI OGAWA: "Function of glutathione in improvement in crop yield and quality", RESEARCH JOURNAL OF FOOD AND AGRICULTURE, vol. 32, no. 3, 1 March 2009 (2009-03-01), pages 44 - 47 * |
| KENJI KOMATSU ET AL.: "Physcomitrella patens ni Okeru Protein Phosphatase 2C o Kaishita ABA Signal Dentatsu no Kaiseki", THE JAPANESE SOCIETY OF PLANT PHYSIOLOGISTS NENKAI YOSHISHU, 15 March 2007 (2007-03-15), pages 131 * |
Cited By (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2012039159A1 (ja) * | 2010-09-22 | 2012-03-29 | 国立大学法人奈良先端科学技術大学院大学 | 塊茎生産能または匍匐枝形成能が野生株に比して向上している匍匐枝形成植物の作製方法、当該方法によって作製された匍匐枝形成植物 |
| JP5787413B2 (ja) * | 2010-09-22 | 2015-09-30 | 国立大学法人 奈良先端科学技術大学院大学 | 塊茎生産能または匍匐枝形成能が野生株に比して向上している匍匐枝形成植物の作製方法、当該方法によって作製された匍匐枝形成植物 |
Also Published As
| Publication number | Publication date |
|---|---|
| US9546376B2 (en) | 2017-01-17 |
| AU2010222106A1 (en) | 2011-11-03 |
| JP5604657B2 (ja) | 2014-10-08 |
| AU2010222106B2 (en) | 2013-08-29 |
| CN102395675A (zh) | 2012-03-28 |
| JP2010207199A (ja) | 2010-09-24 |
| BRPI1009784A2 (pt) | 2015-08-25 |
| US20120005787A1 (en) | 2012-01-05 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP5403628B2 (ja) | 植物のバイオマス量及び/又は種子量を増産させる遺伝子及びその利用方法 | |
| JP5250807B2 (ja) | 植物のバイオマス量及び/又は種子量を増産させる方法、バイオマス量及び/又は種子量を増産できる植物の製造方法 | |
| WO2009110466A1 (ja) | 植物の油脂を増産させる遺伝子及びその利用方法 | |
| JP5604657B2 (ja) | 植物のバイオマス量及び/又は種子量を増産させる遺伝子及びその利用方法 | |
| JP5672004B2 (ja) | 植物のバイオマス量を増産させる遺伝子及びその利用方法 | |
| JP5454086B2 (ja) | 植物に環境ストレス耐性を付与する遺伝子及びその利用方法 | |
| JP5212955B2 (ja) | 植物のバイオマス量を増産させる遺伝子及びその利用方法 | |
| JP5686977B2 (ja) | 植物の油脂生産性を増大させる遺伝子及びその利用方法 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| WWE | Wipo information: entry into national phase |
Ref document number: 201080011392.1 Country of ref document: CN |
|
| 121 | Ep: the epo has been informed by wipo that ep was designated in this application |
Ref document number: 10750845 Country of ref document: EP Kind code of ref document: A1 |
|
| NENP | Non-entry into the national phase |
Ref country code: DE |
|
| WWE | Wipo information: entry into national phase |
Ref document number: 13256190 Country of ref document: US |
|
| ENP | Entry into the national phase |
Ref document number: 2010222106 Country of ref document: AU Date of ref document: 20100310 Kind code of ref document: A |
|
| 122 | Ep: pct application non-entry in european phase |
Ref document number: 10750845 Country of ref document: EP Kind code of ref document: A1 |
|
| REG | Reference to national code |
Ref country code: BR Ref legal event code: B01A Ref document number: PI1009784 Country of ref document: BR |
|
| ENP | Entry into the national phase |
Ref document number: PI1009784 Country of ref document: BR Kind code of ref document: A2 Effective date: 20110912 |