KR20230050336A - 뇌전증을 치료하기 위한 방법과 조성물 - Google Patents
뇌전증을 치료하기 위한 방법과 조성물 Download PDFInfo
- Publication number
- KR20230050336A KR20230050336A KR1020237004876A KR20237004876A KR20230050336A KR 20230050336 A KR20230050336 A KR 20230050336A KR 1020237004876 A KR1020237004876 A KR 1020237004876A KR 20237004876 A KR20237004876 A KR 20237004876A KR 20230050336 A KR20230050336 A KR 20230050336A
- Authority
- KR
- South Korea
- Prior art keywords
- seq
- sequence
- nucleic acid
- sequence identity
- contiguous nucleotides
- Prior art date
Links
- 238000000034 method Methods 0.000 title claims abstract description 113
- 206010015037 epilepsy Diseases 0.000 title claims abstract description 43
- 239000000203 mixture Substances 0.000 title abstract description 34
- 101150006929 GRIK2 gene Proteins 0.000 claims abstract description 317
- 108020004999 messenger RNA Proteins 0.000 claims abstract description 292
- 230000014509 gene expression Effects 0.000 claims abstract description 223
- 201000008914 temporal lobe epilepsy Diseases 0.000 claims abstract description 74
- 102100022758 Glutamate receptor ionotropic, kainate 2 Human genes 0.000 claims abstract description 65
- 101710112360 Glutamate receptor ionotropic, kainate 2 Proteins 0.000 claims abstract description 60
- 230000002401 inhibitory effect Effects 0.000 claims abstract description 17
- 108091032973 (ribonucleotides)n+m Proteins 0.000 claims abstract description 10
- 150000007523 nucleic acids Chemical group 0.000 claims description 608
- 125000003729 nucleotide group Chemical group 0.000 claims description 580
- 239000002773 nucleotide Substances 0.000 claims description 579
- 108091028043 Nucleic acid sequence Proteins 0.000 claims description 481
- 108091034117 Oligonucleotide Proteins 0.000 claims description 384
- 239000000074 antisense oligonucleotide Substances 0.000 claims description 352
- 238000012230 antisense oligonucleotides Methods 0.000 claims description 352
- 102000040430 polynucleotide Human genes 0.000 claims description 312
- 108091033319 polynucleotide Proteins 0.000 claims description 312
- 239000002157 polynucleotide Substances 0.000 claims description 312
- 230000000295 complement effect Effects 0.000 claims description 140
- 239000013598 vector Substances 0.000 claims description 135
- 239000002679 microRNA Substances 0.000 claims description 128
- 108700011259 MicroRNAs Proteins 0.000 claims description 107
- 239000004055 small Interfering RNA Substances 0.000 claims description 93
- 108020004459 Small interfering RNA Proteins 0.000 claims description 87
- 210000004027 cell Anatomy 0.000 claims description 74
- 108090000623 proteins and genes Proteins 0.000 claims description 72
- 108020005065 3' Flanking Region Proteins 0.000 claims description 52
- 108020005029 5' Flanking Region Proteins 0.000 claims description 52
- 210000004565 granule cell Anatomy 0.000 claims description 48
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 claims description 24
- 108091027967 Small hairpin RNA Proteins 0.000 claims description 23
- 102000004169 proteins and genes Human genes 0.000 claims description 22
- 230000027455 binding Effects 0.000 claims description 21
- 230000015572 biosynthetic process Effects 0.000 claims description 21
- 241000282414 Homo sapiens Species 0.000 claims description 19
- 230000008488 polyadenylation Effects 0.000 claims description 19
- 239000013603 viral vector Substances 0.000 claims description 19
- 108020004414 DNA Proteins 0.000 claims description 18
- 108091026890 Coding region Proteins 0.000 claims description 16
- 210000002569 neuron Anatomy 0.000 claims description 14
- 239000008194 pharmaceutical composition Substances 0.000 claims description 14
- 108010006025 bovine growth hormone Proteins 0.000 claims description 13
- 210000004295 hippocampal neuron Anatomy 0.000 claims description 13
- 230000009467 reduction Effects 0.000 claims description 13
- 239000013607 AAV vector Substances 0.000 claims description 12
- WHUUTDBJXJRKMK-VKHMYHEASA-N L-glutamic acid Chemical compound OC(=O)[C@@H](N)CCC(O)=O WHUUTDBJXJRKMK-VKHMYHEASA-N 0.000 claims description 11
- 230000001684 chronic effect Effects 0.000 claims description 11
- 102100021244 Integral membrane protein GPR180 Human genes 0.000 claims description 10
- 239000013604 expression vector Substances 0.000 claims description 10
- 230000000306 recurrent effect Effects 0.000 claims description 10
- 229930195712 glutamate Natural products 0.000 claims description 9
- 108020005345 3' Untranslated Regions Proteins 0.000 claims description 8
- 108020003589 5' Untranslated Regions Proteins 0.000 claims description 8
- 230000010076 replication Effects 0.000 claims description 8
- 102000008214 Glutamate decarboxylase Human genes 0.000 claims description 7
- 108091022930 Glutamate decarboxylase Proteins 0.000 claims description 7
- 241000702423 Adeno-associated virus - 2 Species 0.000 claims description 6
- 102100020802 D(1A) dopamine receptor Human genes 0.000 claims description 6
- 102100020756 D(2) dopamine receptor Human genes 0.000 claims description 6
- 101000931925 Homo sapiens D(1A) dopamine receptor Proteins 0.000 claims description 6
- 101000931901 Homo sapiens D(2) dopamine receptor Proteins 0.000 claims description 6
- 101001092197 Homo sapiens RNA binding protein fox-1 homolog 3 Proteins 0.000 claims description 6
- 102000004866 Microtubule-associated protein 1B Human genes 0.000 claims description 6
- 108090001040 Microtubule-associated protein 1B Proteins 0.000 claims description 6
- 108060005874 Parvalbumin Proteins 0.000 claims description 6
- 102000001675 Parvalbumin Human genes 0.000 claims description 6
- 102000012288 Phosphopyruvate Hydratase Human genes 0.000 claims description 6
- 108010022181 Phosphopyruvate Hydratase Proteins 0.000 claims description 6
- 108010069820 Pro-Opiomelanocortin Proteins 0.000 claims description 6
- 239000000683 Pro-Opiomelanocortin Substances 0.000 claims description 6
- 102100027467 Pro-opiomelanocortin Human genes 0.000 claims description 6
- 102100035530 RNA binding protein fox-1 homolog 3 Human genes 0.000 claims description 6
- 102000005157 Somatostatin Human genes 0.000 claims description 6
- 108010056088 Somatostatin Proteins 0.000 claims description 6
- 102000002248 Thyroxine-Binding Globulin Human genes 0.000 claims description 6
- 108010000259 Thyroxine-Binding Globulin Proteins 0.000 claims description 6
- 108010003205 Vasoactive Intestinal Peptide Proteins 0.000 claims description 6
- 230000003042 antagnostic effect Effects 0.000 claims description 6
- VBUWHHLIZKOSMS-RIWXPGAOSA-N invicorp Chemical compound C([C@@H](C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CO)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CC(N)=O)C(O)=O)NC(=O)[C@H](CCCCN)NC(=O)[C@H](CCCCN)NC(=O)[C@@H](NC(=O)[C@H](C)NC(=O)[C@H](CCSC)NC(=O)[C@H](CCC(N)=O)NC(=O)[C@H](CCCCN)NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@@H](NC(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(O)=O)NC(=O)[C@@H](NC(=O)[C@H](CC=1C=CC=CC=1)NC(=O)[C@@H](NC(=O)[C@H](C)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CC=1NC=NC=1)C(C)C)[C@@H](C)O)[C@@H](C)O)C(C)C)C1=CC=C(O)C=C1 VBUWHHLIZKOSMS-RIWXPGAOSA-N 0.000 claims description 6
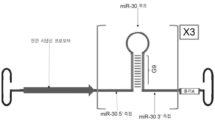
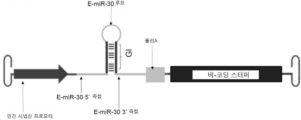
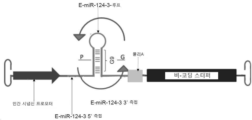
- 108091048120 miR-124-3 stem-loop Proteins 0.000 claims description 6
- 239000000546 pharmaceutical excipient Substances 0.000 claims description 6
- NHXLMOGPVYXJNR-ATOGVRKGSA-N somatostatin Chemical compound C([C@H]1C(=O)N[C@H](C(N[C@@H](CO)C(=O)N[C@@H](CSSC[C@@H](C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CC=2C=CC=CC=2)C(=O)N[C@@H](CC=2C=CC=CC=2)C(=O)N[C@@H](CC=2C3=CC=CC=C3NC=2)C(=O)N[C@@H](CCCCN)C(=O)N[C@H](C(=O)N1)[C@@H](C)O)NC(=O)CNC(=O)[C@H](C)N)C(O)=O)=O)[C@H](O)C)C1=CC=CC=C1 NHXLMOGPVYXJNR-ATOGVRKGSA-N 0.000 claims description 6
- 229960000553 somatostatin Drugs 0.000 claims description 6
- 125000006850 spacer group Chemical group 0.000 claims description 6
- XUIIKFGFIJCVMT-UHFFFAOYSA-N thyroxine-binding globulin Natural products IC1=CC(CC([NH3+])C([O-])=O)=CC(I)=C1OC1=CC(I)=C(O)C(I)=C1 XUIIKFGFIJCVMT-UHFFFAOYSA-N 0.000 claims description 6
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 claims description 5
- 241000702421 Dependoparvovirus Species 0.000 claims description 5
- 108700028146 Genetic Enhancer Elements Proteins 0.000 claims description 5
- 108091026898 Leader sequence (mRNA) Proteins 0.000 claims description 5
- 102000001435 Synapsin Human genes 0.000 claims description 5
- 108050009621 Synapsin Proteins 0.000 claims description 5
- 102000001763 Vesicular Glutamate Transport Proteins Human genes 0.000 claims description 5
- 108010040170 Vesicular Glutamate Transport Proteins Proteins 0.000 claims description 5
- 239000000835 fiber Substances 0.000 claims description 5
- 102000007592 Apolipoproteins Human genes 0.000 claims description 4
- 108010071619 Apolipoproteins Proteins 0.000 claims description 4
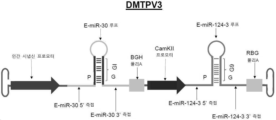
- 101710116137 Calcium/calmodulin-dependent protein kinase II Proteins 0.000 claims description 4
- 102100025881 Complement C1q-like protein 2 Human genes 0.000 claims description 4
- 102000053602 DNA Human genes 0.000 claims description 4
- 101000933639 Homo sapiens Complement C1q-like protein 2 Proteins 0.000 claims description 4
- 101710151321 Melanostatin Proteins 0.000 claims description 4
- 108091033773 MiR-155 Proteins 0.000 claims description 4
- 102400000064 Neuropeptide Y Human genes 0.000 claims description 4
- 102000010292 Peptide Elongation Factor 1 Human genes 0.000 claims description 4
- 108010077524 Peptide Elongation Factor 1 Proteins 0.000 claims description 4
- 102000010780 Platelet-Derived Growth Factor Human genes 0.000 claims description 4
- 108010038512 Platelet-Derived Growth Factor Proteins 0.000 claims description 4
- 108091023045 Untranslated Region Proteins 0.000 claims description 4
- 102000015395 alpha 1-Antitrypsin Human genes 0.000 claims description 4
- 108010050122 alpha 1-Antitrypsin Proteins 0.000 claims description 4
- 229940024142 alpha 1-antitrypsin Drugs 0.000 claims description 4
- 230000001580 bacterial effect Effects 0.000 claims description 4
- 108010085933 diguanylate cyclase Proteins 0.000 claims description 4
- 241001493065 dsRNA viruses Species 0.000 claims description 4
- 108091065218 miR-218-1 stem-loop Proteins 0.000 claims description 4
- URPYMXQQVHTUDU-OFGSCBOVSA-N nucleopeptide y Chemical compound C([C@@H](C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(N)=O)NC(=O)[C@H](CC=1NC=NC=1)NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](C)NC(=O)[C@H](CO)NC(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@H](C)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](C)NC(=O)[C@H]1N(CCC1)C(=O)[C@H](C)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)CNC(=O)[C@H]1N(CCC1)C(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H]1N(CCC1)C(=O)[C@H](CCCCN)NC(=O)[C@H](CO)NC(=O)[C@H]1N(CCC1)C(=O)[C@@H](N)CC=1C=CC(O)=CC=1)C1=CC=C(O)C=C1 URPYMXQQVHTUDU-OFGSCBOVSA-N 0.000 claims description 4
- 208000001654 Drug Resistant Epilepsy Diseases 0.000 claims description 3
- 102100021519 Hemoglobin subunit beta Human genes 0.000 claims description 3
- 108091005904 Hemoglobin subunit beta Proteins 0.000 claims description 3
- 241000283973 Oryctolagus cuniculus Species 0.000 claims description 3
- 102000004243 Tubulin Human genes 0.000 claims description 3
- 108090000704 Tubulin Proteins 0.000 claims description 3
- 230000002950 deficient Effects 0.000 claims description 3
- 239000003085 diluting agent Substances 0.000 claims description 3
- 241000710929 Alphavirus Species 0.000 claims description 2
- 241000178270 Canarypox virus Species 0.000 claims description 2
- 241000711573 Coronaviridae Species 0.000 claims description 2
- 241000701022 Cytomegalovirus Species 0.000 claims description 2
- 102000004190 Enzymes Human genes 0.000 claims description 2
- 108090000790 Enzymes Proteins 0.000 claims description 2
- 241000700662 Fowlpox virus Species 0.000 claims description 2
- 241000701044 Human gammaherpesvirus 4 Species 0.000 claims description 2
- 241000125945 Protoparvovirus Species 0.000 claims description 2
- 210000003050 axon Anatomy 0.000 claims description 2
- 241000701161 unidentified adenovirus Species 0.000 claims description 2
- 241001529453 unidentified herpesvirus Species 0.000 claims description 2
- 241001430294 unidentified retrovirus Species 0.000 claims description 2
- 102000055135 Vasoactive Intestinal Peptide Human genes 0.000 claims 2
- 241000450599 DNA viruses Species 0.000 claims 1
- 241000709664 Picornaviridae Species 0.000 claims 1
- 238000006114 decarboxylation reaction Methods 0.000 claims 1
- 230000001037 epileptic effect Effects 0.000 abstract description 29
- 230000008685 targeting Effects 0.000 abstract description 8
- 230000001537 neural effect Effects 0.000 abstract description 6
- 238000001415 gene therapy Methods 0.000 abstract description 2
- 239000000556 agonist Substances 0.000 description 81
- 230000000692 anti-sense effect Effects 0.000 description 59
- 101000575685 Homo sapiens Synembryn-B Proteins 0.000 description 48
- 102100026014 Synembryn-B Human genes 0.000 description 48
- 102000039446 nucleic acids Human genes 0.000 description 45
- 108020004707 nucleic acids Proteins 0.000 description 45
- 238000003197 gene knockdown Methods 0.000 description 39
- 238000011282 treatment Methods 0.000 description 37
- 241000699670 Mus sp. Species 0.000 description 36
- 239000002585 base Substances 0.000 description 31
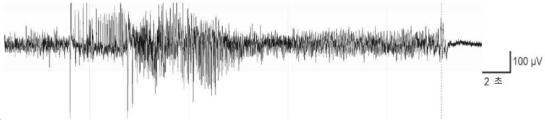
- 206010010904 Convulsion Diseases 0.000 description 27
- 108010069902 Kainic Acid Receptors Proteins 0.000 description 26
- 102000000079 Kainic Acid Receptors Human genes 0.000 description 26
- 102000004657 Calcium-Calmodulin-Dependent Protein Kinase Type 2 Human genes 0.000 description 23
- 108010003721 Calcium-Calmodulin-Dependent Protein Kinase Type 2 Proteins 0.000 description 23
- 230000000875 corresponding effect Effects 0.000 description 22
- 210000004556 brain Anatomy 0.000 description 21
- 150000001413 amino acids Chemical group 0.000 description 20
- 235000018102 proteins Nutrition 0.000 description 20
- -1 at least 5 Chemical compound 0.000 description 18
- 239000005090 green fluorescent protein Substances 0.000 description 18
- 108010043121 Green Fluorescent Proteins Proteins 0.000 description 17
- 102000004144 Green Fluorescent Proteins Human genes 0.000 description 17
- 230000000971 hippocampal effect Effects 0.000 description 17
- 239000003795 chemical substances by application Substances 0.000 description 15
- 201000010099 disease Diseases 0.000 description 14
- 239000013612 plasmid Substances 0.000 description 14
- 230000035897 transcription Effects 0.000 description 14
- 238000013518 transcription Methods 0.000 description 14
- 238000002347 injection Methods 0.000 description 13
- 239000007924 injection Substances 0.000 description 13
- 102100022761 Glutamate receptor ionotropic, kainate 5 Human genes 0.000 description 12
- 101710112356 Glutamate receptor ionotropic, kainate 5 Proteins 0.000 description 12
- 235000001014 amino acid Nutrition 0.000 description 12
- 230000000694 effects Effects 0.000 description 12
- 238000012545 processing Methods 0.000 description 12
- 230000014616 translation Effects 0.000 description 12
- 101100272893 Mus musculus C1ql2 gene Proteins 0.000 description 11
- 229940024606 amino acid Drugs 0.000 description 11
- 239000003814 drug Substances 0.000 description 11
- 230000009977 dual effect Effects 0.000 description 11
- 239000003112 inhibitor Substances 0.000 description 11
- 230000001105 regulatory effect Effects 0.000 description 11
- 238000013519 translation Methods 0.000 description 11
- 108020000948 Antisense Oligonucleotides Proteins 0.000 description 10
- 210000001947 dentate gyrus Anatomy 0.000 description 10
- 210000001320 hippocampus Anatomy 0.000 description 10
- 108091070501 miRNA Proteins 0.000 description 10
- 238000006467 substitution reaction Methods 0.000 description 10
- 208000011580 syndromic disease Diseases 0.000 description 10
- QCHFTSOMWOSFHM-WPRPVWTQSA-N (+)-Pilocarpine Chemical compound C1OC(=O)[C@@H](CC)[C@H]1CC1=CN=CN1C QCHFTSOMWOSFHM-WPRPVWTQSA-N 0.000 description 9
- 230000001404 mediated effect Effects 0.000 description 9
- 239000000047 product Substances 0.000 description 9
- 230000002829 reductive effect Effects 0.000 description 9
- 238000012360 testing method Methods 0.000 description 9
- 238000012228 RNA interference-mediated gene silencing Methods 0.000 description 8
- IQFYYKKMVGJFEH-XLPZGREQSA-N Thymidine Chemical compound O=C1NC(=O)C(C)=CN1[C@@H]1O[C@H](CO)[C@@H](O)C1 IQFYYKKMVGJFEH-XLPZGREQSA-N 0.000 description 8
- DRTQHJPVMGBUCF-XVFCMESISA-N Uridine Chemical compound O[C@@H]1[C@H](O)[C@@H](CO)O[C@H]1N1C(=O)NC(=O)C=C1 DRTQHJPVMGBUCF-XVFCMESISA-N 0.000 description 8
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 description 8
- 239000002253 acid Substances 0.000 description 8
- 150000001875 compounds Chemical class 0.000 description 8
- 208000035475 disorder Diseases 0.000 description 8
- 229940079593 drug Drugs 0.000 description 8
- 230000009368 gene silencing by RNA Effects 0.000 description 8
- 229940049906 glutamate Drugs 0.000 description 8
- 238000003468 luciferase reporter gene assay Methods 0.000 description 8
- 210000003478 temporal lobe Anatomy 0.000 description 8
- 210000001519 tissue Anatomy 0.000 description 8
- 102000040650 (ribonucleotides)n+m Human genes 0.000 description 7
- 208000002267 Anti-neutrophil cytoplasmic antibody-associated vasculitis Diseases 0.000 description 7
- 101001069749 Homo sapiens Prospero homeobox protein 1 Proteins 0.000 description 7
- 102100033880 Prospero homeobox protein 1 Human genes 0.000 description 7
- 238000000338 in vitro Methods 0.000 description 7
- 238000001727 in vivo Methods 0.000 description 7
- 210000004263 induced pluripotent stem cell Anatomy 0.000 description 7
- 230000005764 inhibitory process Effects 0.000 description 7
- 238000012423 maintenance Methods 0.000 description 7
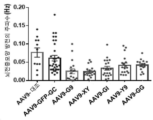
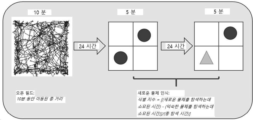
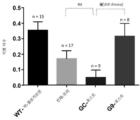
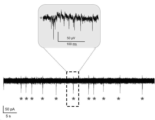
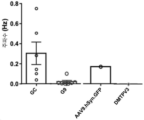
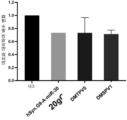
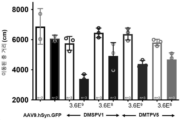
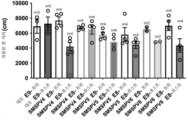
- 238000012346 open field test Methods 0.000 description 7
- 150000003839 salts Chemical class 0.000 description 7
- 238000011269 treatment regimen Methods 0.000 description 7
- 102000018899 Glutamate Receptors Human genes 0.000 description 6
- 108010027915 Glutamate Receptors Proteins 0.000 description 6
- 241001529936 Murinae Species 0.000 description 6
- QCHFTSOMWOSFHM-UHFFFAOYSA-N SJ000285536 Natural products C1OC(=O)C(CC)C1CC1=CN=CN1C QCHFTSOMWOSFHM-UHFFFAOYSA-N 0.000 description 6
- 108091036066 Three prime untranslated region Proteins 0.000 description 6
- 239000002299 complementary DNA Substances 0.000 description 6
- 230000007423 decrease Effects 0.000 description 6
- 229910052739 hydrogen Inorganic materials 0.000 description 6
- 229960001416 pilocarpine Drugs 0.000 description 6
- 102000005962 receptors Human genes 0.000 description 6
- 108020003175 receptors Proteins 0.000 description 6
- 102100023387 Endoribonuclease Dicer Human genes 0.000 description 5
- 108700024394 Exon Proteins 0.000 description 5
- 101000834253 Gallus gallus Actin, cytoplasmic 1 Proteins 0.000 description 5
- 101000907904 Homo sapiens Endoribonuclease Dicer Proteins 0.000 description 5
- 101000903346 Homo sapiens Glutamate receptor ionotropic, kainate 2 Proteins 0.000 description 5
- 230000015556 catabolic process Effects 0.000 description 5
- 230000008859 change Effects 0.000 description 5
- 238000006731 degradation reaction Methods 0.000 description 5
- 238000010586 diagram Methods 0.000 description 5
- 230000006870 function Effects 0.000 description 5
- 239000001257 hydrogen Substances 0.000 description 5
- 230000002296 hyperlocomotor Effects 0.000 description 5
- 230000006698 induction Effects 0.000 description 5
- 238000004806 packaging method and process Methods 0.000 description 5
- 108090000765 processed proteins & peptides Proteins 0.000 description 5
- 230000002269 spontaneous effect Effects 0.000 description 5
- 210000000225 synapse Anatomy 0.000 description 5
- DWRXFEITVBNRMK-UHFFFAOYSA-N Beta-D-1-Arabinofuranosylthymine Natural products O=C1NC(=O)C(C)=CN1C1C(O)C(O)C(CO)O1 DWRXFEITVBNRMK-UHFFFAOYSA-N 0.000 description 4
- 102100022197 Glutamate receptor ionotropic, kainate 1 Human genes 0.000 description 4
- 101710112359 Glutamate receptor ionotropic, kainate 1 Proteins 0.000 description 4
- 102100022765 Glutamate receptor ionotropic, kainate 4 Human genes 0.000 description 4
- 101710112358 Glutamate receptor ionotropic, kainate 4 Proteins 0.000 description 4
- 241000124008 Mammalia Species 0.000 description 4
- 208000036572 Myoclonic epilepsy Diseases 0.000 description 4
- 206010061334 Partial seizures Diseases 0.000 description 4
- 102400000015 Vasoactive intestinal peptide Human genes 0.000 description 4
- 241000700605 Viruses Species 0.000 description 4
- OIRDTQYFTABQOQ-KQYNXXCUSA-N adenosine Chemical compound C1=NC=2C(N)=NC=NC=2N1[C@@H]1O[C@H](CO)[C@@H](O)[C@H]1O OIRDTQYFTABQOQ-KQYNXXCUSA-N 0.000 description 4
- 230000000712 assembly Effects 0.000 description 4
- 238000000429 assembly Methods 0.000 description 4
- IQFYYKKMVGJFEH-UHFFFAOYSA-N beta-L-thymidine Natural products O=C1NC(=O)C(C)=CN1C1OC(CO)C(O)C1 IQFYYKKMVGJFEH-UHFFFAOYSA-N 0.000 description 4
- DRTQHJPVMGBUCF-PSQAKQOGSA-N beta-L-uridine Natural products O[C@H]1[C@@H](O)[C@H](CO)O[C@@H]1N1C(=O)NC(=O)C=C1 DRTQHJPVMGBUCF-PSQAKQOGSA-N 0.000 description 4
- 210000003169 central nervous system Anatomy 0.000 description 4
- 238000003776 cleavage reaction Methods 0.000 description 4
- 201000009028 early myoclonic encephalopathy Diseases 0.000 description 4
- 239000003623 enhancer Substances 0.000 description 4
- 230000030279 gene silencing Effects 0.000 description 4
- 238000009396 hybridization Methods 0.000 description 4
- 230000001965 increasing effect Effects 0.000 description 4
- 230000003902 lesion Effects 0.000 description 4
- 238000004519 manufacturing process Methods 0.000 description 4
- 230000037361 pathway Effects 0.000 description 4
- 229920001184 polypeptide Polymers 0.000 description 4
- 239000002243 precursor Substances 0.000 description 4
- 102000004196 processed proteins & peptides Human genes 0.000 description 4
- 238000003753 real-time PCR Methods 0.000 description 4
- 230000008521 reorganization Effects 0.000 description 4
- 230000007017 scission Effects 0.000 description 4
- 230000001225 therapeutic effect Effects 0.000 description 4
- 229940104230 thymidine Drugs 0.000 description 4
- 238000010361 transduction Methods 0.000 description 4
- 230000026683 transduction Effects 0.000 description 4
- DRTQHJPVMGBUCF-UHFFFAOYSA-N uracil arabinoside Natural products OC1C(O)C(CO)OC1N1C(=O)NC(=O)C=C1 DRTQHJPVMGBUCF-UHFFFAOYSA-N 0.000 description 4
- 229940045145 uridine Drugs 0.000 description 4
- 241001655883 Adeno-associated virus - 1 Species 0.000 description 3
- 241000202702 Adeno-associated virus - 3 Species 0.000 description 3
- 241000580270 Adeno-associated virus - 4 Species 0.000 description 3
- 241001634120 Adeno-associated virus - 5 Species 0.000 description 3
- 241000972680 Adeno-associated virus - 6 Species 0.000 description 3
- 241001164823 Adeno-associated virus - 7 Species 0.000 description 3
- 241001164825 Adeno-associated virus - 8 Species 0.000 description 3
- 241000649045 Adeno-associated virus 10 Species 0.000 description 3
- 241000649046 Adeno-associated virus 11 Species 0.000 description 3
- 241000649047 Adeno-associated virus 12 Species 0.000 description 3
- 241000300529 Adeno-associated virus 13 Species 0.000 description 3
- 241000958487 Adeno-associated virus 3B Species 0.000 description 3
- 108020004705 Codon Proteins 0.000 description 3
- 102100035426 DnaJ homolog subfamily B member 7 Human genes 0.000 description 3
- 101100285903 Drosophila melanogaster Hsc70-2 gene Proteins 0.000 description 3
- 101100178718 Drosophila melanogaster Hsc70-4 gene Proteins 0.000 description 3
- 101100178723 Drosophila melanogaster Hsc70-5 gene Proteins 0.000 description 3
- 208000002091 Febrile Seizures Diseases 0.000 description 3
- 102100022767 Glutamate receptor ionotropic, kainate 3 Human genes 0.000 description 3
- 101710112357 Glutamate receptor ionotropic, kainate 3 Proteins 0.000 description 3
- 101000804114 Homo sapiens DnaJ homolog subfamily B member 7 Proteins 0.000 description 3
- 101150090950 Hsc70-1 gene Proteins 0.000 description 3
- VLSMHEGGTFMBBZ-OOZYFLPDSA-M Kainate Chemical compound CC(=C)[C@H]1C[NH2+][C@H](C([O-])=O)[C@H]1CC([O-])=O VLSMHEGGTFMBBZ-OOZYFLPDSA-M 0.000 description 3
- 108091027974 Mature messenger RNA Proteins 0.000 description 3
- 101100150366 Schizosaccharomyces pombe (strain 972 / ATCC 24843) sks2 gene Proteins 0.000 description 3
- 241000700584 Simplexvirus Species 0.000 description 3
- 108091081024 Start codon Proteins 0.000 description 3
- 208000006011 Stroke Diseases 0.000 description 3
- ZMANZCXQSJIPKH-UHFFFAOYSA-N Triethylamine Chemical compound CCN(CC)CC ZMANZCXQSJIPKH-UHFFFAOYSA-N 0.000 description 3
- 150000007513 acids Chemical class 0.000 description 3
- 230000001154 acute effect Effects 0.000 description 3
- 230000007177 brain activity Effects 0.000 description 3
- 230000001413 cellular effect Effects 0.000 description 3
- KRKNYBCHXYNGOX-UHFFFAOYSA-N citric acid Chemical compound OC(=O)CC(O)(C(O)=O)CC(O)=O KRKNYBCHXYNGOX-UHFFFAOYSA-N 0.000 description 3
- 239000003184 complementary RNA Substances 0.000 description 3
- 210000003618 cortical neuron Anatomy 0.000 description 3
- 238000010864 dual luciferase reporter gene assay Methods 0.000 description 3
- 206010014599 encephalitis Diseases 0.000 description 3
- 238000005516 engineering process Methods 0.000 description 3
- 230000002964 excitative effect Effects 0.000 description 3
- 208000015181 infectious disease Diseases 0.000 description 3
- 230000003834 intracellular effect Effects 0.000 description 3
- 238000002372 labelling Methods 0.000 description 3
- 150000002632 lipids Chemical class 0.000 description 3
- 239000000463 material Substances 0.000 description 3
- 231100000252 nontoxic Toxicity 0.000 description 3
- 230000003000 nontoxic effect Effects 0.000 description 3
- 125000003835 nucleoside group Chemical group 0.000 description 3
- 239000012071 phase Substances 0.000 description 3
- 229940095606 pilocar Drugs 0.000 description 3
- 229920000036 polyvinylpyrrolidone Polymers 0.000 description 3
- 235000013855 polyvinylpyrrolidone Nutrition 0.000 description 3
- 229910052700 potassium Inorganic materials 0.000 description 3
- 230000008569 process Effects 0.000 description 3
- 230000009712 regulation of translation Effects 0.000 description 3
- 239000000126 substance Substances 0.000 description 3
- 230000002123 temporal effect Effects 0.000 description 3
- 238000002560 therapeutic procedure Methods 0.000 description 3
- GVJHHUAWPYXKBD-UHFFFAOYSA-N (±)-α-Tocopherol Chemical compound OC1=C(C)C(C)=C2OC(CCCC(C)CCCC(C)CCCC(C)C)(C)CCC2=C1C GVJHHUAWPYXKBD-UHFFFAOYSA-N 0.000 description 2
- QGZKDVFQNNGYKY-UHFFFAOYSA-O Ammonium Chemical compound [NH4+] QGZKDVFQNNGYKY-UHFFFAOYSA-O 0.000 description 2
- 108020004491 Antisense DNA Proteins 0.000 description 2
- 108020005544 Antisense RNA Proteins 0.000 description 2
- 239000004322 Butylated hydroxytoluene Substances 0.000 description 2
- NLZUEZXRPGMBCV-UHFFFAOYSA-N Butylhydroxytoluene Chemical compound CC1=CC(C(C)(C)C)=C(O)C(C(C)(C)C)=C1 NLZUEZXRPGMBCV-UHFFFAOYSA-N 0.000 description 2
- 239000002126 C01EB10 - Adenosine Substances 0.000 description 2
- VTYYLEPIZMXCLO-UHFFFAOYSA-L Calcium carbonate Chemical compound [Ca+2].[O-]C([O-])=O VTYYLEPIZMXCLO-UHFFFAOYSA-L 0.000 description 2
- 108020004394 Complementary RNA Proteins 0.000 description 2
- 108090000626 DNA-directed RNA polymerases Proteins 0.000 description 2
- 102000004163 DNA-directed RNA polymerases Human genes 0.000 description 2
- ROSDSFDQCJNGOL-UHFFFAOYSA-N Dimethylamine Chemical compound CNC ROSDSFDQCJNGOL-UHFFFAOYSA-N 0.000 description 2
- 238000002965 ELISA Methods 0.000 description 2
- QUSNBJAOOMFDIB-UHFFFAOYSA-N Ethylamine Chemical compound CCN QUSNBJAOOMFDIB-UHFFFAOYSA-N 0.000 description 2
- WHUUTDBJXJRKMK-UHFFFAOYSA-N Glutamic acid Natural products OC(=O)C(N)CCC(O)=O WHUUTDBJXJRKMK-UHFFFAOYSA-N 0.000 description 2
- NYHBQMYGNKIUIF-UUOKFMHZSA-N Guanosine Chemical compound C1=NC=2C(=O)NC(N)=NC=2N1[C@@H]1O[C@H](CO)[C@@H](O)[C@H]1O NYHBQMYGNKIUIF-UUOKFMHZSA-N 0.000 description 2
- 101000838463 Homo sapiens Tubulin alpha-1A chain Proteins 0.000 description 2
- DGAQECJNVWCQMB-PUAWFVPOSA-M Ilexoside XXIX Chemical compound C[C@@H]1CC[C@@]2(CC[C@@]3(C(=CC[C@H]4[C@]3(CC[C@@H]5[C@@]4(CC[C@@H](C5(C)C)OS(=O)(=O)[O-])C)C)[C@@H]2[C@]1(C)O)C)C(=O)O[C@H]6[C@@H]([C@H]([C@@H]([C@H](O6)CO)O)O)O.[Na+] DGAQECJNVWCQMB-PUAWFVPOSA-M 0.000 description 2
- 206010071082 Juvenile myoclonic epilepsy Diseases 0.000 description 2
- 241000713666 Lentivirus Species 0.000 description 2
- 102000004086 Ligand-Gated Ion Channels Human genes 0.000 description 2
- 108090000543 Ligand-Gated Ion Channels Proteins 0.000 description 2
- 201000009906 Meningitis Diseases 0.000 description 2
- 241001465754 Metazoa Species 0.000 description 2
- BAVYZALUXZFZLV-UHFFFAOYSA-N Methylamine Chemical compound NC BAVYZALUXZFZLV-UHFFFAOYSA-N 0.000 description 2
- 241000699666 Mus <mouse, genus> Species 0.000 description 2
- HOKKHZGPKSLGJE-GSVOUGTGSA-N N-Methyl-D-aspartic acid Chemical compound CN[C@@H](C(O)=O)CC(O)=O HOKKHZGPKSLGJE-GSVOUGTGSA-N 0.000 description 2
- WHNWPMSKXPGLAX-UHFFFAOYSA-N N-Vinyl-2-pyrrolidone Chemical compound C=CN1CCCC1=O WHNWPMSKXPGLAX-UHFFFAOYSA-N 0.000 description 2
- 208000012902 Nervous system disease Diseases 0.000 description 2
- OAICVXFJPJFONN-UHFFFAOYSA-N Phosphorus Chemical compound [P] OAICVXFJPJFONN-UHFFFAOYSA-N 0.000 description 2
- ZLMJMSJWJFRBEC-UHFFFAOYSA-N Potassium Chemical compound [K] ZLMJMSJWJFRBEC-UHFFFAOYSA-N 0.000 description 2
- 208000033063 Progressive myoclonic epilepsy Diseases 0.000 description 2
- 230000026279 RNA modification Effects 0.000 description 2
- 102000000574 RNA-Induced Silencing Complex Human genes 0.000 description 2
- 108010016790 RNA-Induced Silencing Complex Proteins 0.000 description 2
- 241000700159 Rattus Species 0.000 description 2
- VYGQUTWHTHXGQB-FFHKNEKCSA-N Retinol Palmitate Chemical compound CCCCCCCCCCCCCCCC(=O)OC\C=C(/C)\C=C\C=C(/C)\C=C\C1=C(C)CCCC1(C)C VYGQUTWHTHXGQB-FFHKNEKCSA-N 0.000 description 2
- 208000034189 Sclerosis Diseases 0.000 description 2
- VYPSYNLAJGMNEJ-UHFFFAOYSA-N Silicium dioxide Chemical compound O=[Si]=O VYPSYNLAJGMNEJ-UHFFFAOYSA-N 0.000 description 2
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 2
- GWEVSGVZZGPLCZ-UHFFFAOYSA-N Titan oxide Chemical compound O=[Ti]=O GWEVSGVZZGPLCZ-UHFFFAOYSA-N 0.000 description 2
- 208000030886 Traumatic Brain injury Diseases 0.000 description 2
- 102100028968 Tubulin alpha-1A chain Human genes 0.000 description 2
- 230000001594 aberrant effect Effects 0.000 description 2
- 229960005305 adenosine Drugs 0.000 description 2
- 229910052784 alkaline earth metal Inorganic materials 0.000 description 2
- 238000004458 analytical method Methods 0.000 description 2
- 239000003816 antisense DNA Substances 0.000 description 2
- 208000008233 autosomal dominant nocturnal frontal lobe epilepsy Diseases 0.000 description 2
- 230000009286 beneficial effect Effects 0.000 description 2
- 230000008901 benefit Effects 0.000 description 2
- 235000010354 butylated hydroxytoluene Nutrition 0.000 description 2
- 229940095259 butylated hydroxytoluene Drugs 0.000 description 2
- 238000004422 calculation algorithm Methods 0.000 description 2
- 238000006243 chemical reaction Methods 0.000 description 2
- 238000000576 coating method Methods 0.000 description 2
- 238000007796 conventional method Methods 0.000 description 2
- 230000002596 correlated effect Effects 0.000 description 2
- 230000001186 cumulative effect Effects 0.000 description 2
- OPTASPLRGRRNAP-UHFFFAOYSA-N cytosine Chemical compound NC=1C=CNC(=O)N=1 OPTASPLRGRRNAP-UHFFFAOYSA-N 0.000 description 2
- 210000000172 cytosol Anatomy 0.000 description 2
- 230000006378 damage Effects 0.000 description 2
- 238000012217 deletion Methods 0.000 description 2
- 230000037430 deletion Effects 0.000 description 2
- MOTZDAYCYVMXPC-UHFFFAOYSA-N dodecyl hydrogen sulfate Chemical compound CCCCCCCCCCCCOS(O)(=O)=O MOTZDAYCYVMXPC-UHFFFAOYSA-N 0.000 description 2
- 229940043264 dodecyl sulfate Drugs 0.000 description 2
- 239000002552 dosage form Substances 0.000 description 2
- 231100000673 dose–response relationship Toxicity 0.000 description 2
- 230000008579 epileptogenesis Effects 0.000 description 2
- 201000007186 focal epilepsy Diseases 0.000 description 2
- 239000000499 gel Substances 0.000 description 2
- 230000002068 genetic effect Effects 0.000 description 2
- 229960002989 glutamic acid Drugs 0.000 description 2
- 235000013922 glutamic acid Nutrition 0.000 description 2
- 239000004220 glutamic acid Substances 0.000 description 2
- UYTPUPDQBNUYGX-UHFFFAOYSA-N guanine Chemical compound O=C1NC(N)=NC2=C1N=CN2 UYTPUPDQBNUYGX-UHFFFAOYSA-N 0.000 description 2
- 230000009524 hypoxic brain injury Effects 0.000 description 2
- 208000034287 idiopathic generalized susceptibility to 7 epilepsy Diseases 0.000 description 2
- 208000014674 injury Diseases 0.000 description 2
- 238000003780 insertion Methods 0.000 description 2
- 230000037431 insertion Effects 0.000 description 2
- 230000010354 integration Effects 0.000 description 2
- 230000002109 interictal effect Effects 0.000 description 2
- 238000007913 intrathecal administration Methods 0.000 description 2
- 238000001990 intravenous administration Methods 0.000 description 2
- 238000010253 intravenous injection Methods 0.000 description 2
- 238000007914 intraventricular administration Methods 0.000 description 2
- 125000005647 linker group Chemical group 0.000 description 2
- 230000033001 locomotion Effects 0.000 description 2
- 239000000314 lubricant Substances 0.000 description 2
- HQKMJHAJHXVSDF-UHFFFAOYSA-L magnesium stearate Chemical compound [Mg+2].CCCCCCCCCCCCCCCCCC([O-])=O.CCCCCCCCCCCCCCCCCC([O-])=O HQKMJHAJHXVSDF-UHFFFAOYSA-L 0.000 description 2
- 230000011987 methylation Effects 0.000 description 2
- 238000007069 methylation reaction Methods 0.000 description 2
- LXCFILQKKLGQFO-UHFFFAOYSA-N methylparaben Chemical compound COC(=O)C1=CC=C(O)C=C1 LXCFILQKKLGQFO-UHFFFAOYSA-N 0.000 description 2
- 150000007522 mineralic acids Chemical class 0.000 description 2
- 230000035772 mutation Effects 0.000 description 2
- 230000002151 myoclonic effect Effects 0.000 description 2
- 210000000478 neocortex Anatomy 0.000 description 2
- 208000018389 neoplasm of cerebral hemisphere Diseases 0.000 description 2
- 230000016273 neuron death Effects 0.000 description 2
- QIQXTHQIDYTFRH-UHFFFAOYSA-N octadecanoic acid Chemical compound CCCCCCCCCCCCCCCCCC(O)=O QIQXTHQIDYTFRH-UHFFFAOYSA-N 0.000 description 2
- 150000007524 organic acids Chemical class 0.000 description 2
- 235000005985 organic acids Nutrition 0.000 description 2
- 230000001314 paroxysmal effect Effects 0.000 description 2
- 230000001991 pathophysiological effect Effects 0.000 description 2
- 230000007310 pathophysiology Effects 0.000 description 2
- 229910052698 phosphorus Inorganic materials 0.000 description 2
- 239000011574 phosphorus Substances 0.000 description 2
- 239000001267 polyvinylpyrrolidone Substances 0.000 description 2
- 239000011591 potassium Substances 0.000 description 2
- 230000002265 prevention Effects 0.000 description 2
- 201000001204 progressive myoclonus epilepsy Diseases 0.000 description 2
- 238000011321 prophylaxis Methods 0.000 description 2
- QELSKZZBTMNZEB-UHFFFAOYSA-N propylparaben Chemical compound CCCOC(=O)C1=CC=C(O)C=C1 QELSKZZBTMNZEB-UHFFFAOYSA-N 0.000 description 2
- 238000011002 quantification Methods 0.000 description 2
- 230000004044 response Effects 0.000 description 2
- 229910052708 sodium Inorganic materials 0.000 description 2
- 239000011734 sodium Substances 0.000 description 2
- 230000001629 suppression Effects 0.000 description 2
- 208000024891 symptom Diseases 0.000 description 2
- 231100000419 toxicity Toxicity 0.000 description 2
- 230000001988 toxicity Effects 0.000 description 2
- VZCYOOQTPOCHFL-UHFFFAOYSA-N trans-butenedioic acid Natural products OC(=O)C=CC(O)=O VZCYOOQTPOCHFL-UHFFFAOYSA-N 0.000 description 2
- 230000009529 traumatic brain injury Effects 0.000 description 2
- GETQZCLCWQTVFV-UHFFFAOYSA-N trimethylamine Chemical compound CN(C)C GETQZCLCWQTVFV-UHFFFAOYSA-N 0.000 description 2
- 238000011144 upstream manufacturing Methods 0.000 description 2
- 210000002845 virion Anatomy 0.000 description 2
- 230000003612 virological effect Effects 0.000 description 2
- 238000001262 western blot Methods 0.000 description 2
- LNAZSHAWQACDHT-XIYTZBAFSA-N (2r,3r,4s,5r,6s)-4,5-dimethoxy-2-(methoxymethyl)-3-[(2s,3r,4s,5r,6r)-3,4,5-trimethoxy-6-(methoxymethyl)oxan-2-yl]oxy-6-[(2r,3r,4s,5r,6r)-4,5,6-trimethoxy-2-(methoxymethyl)oxan-3-yl]oxyoxane Chemical compound CO[C@@H]1[C@@H](OC)[C@H](OC)[C@@H](COC)O[C@H]1O[C@H]1[C@H](OC)[C@@H](OC)[C@H](O[C@H]2[C@@H]([C@@H](OC)[C@H](OC)O[C@@H]2COC)OC)O[C@@H]1COC LNAZSHAWQACDHT-XIYTZBAFSA-N 0.000 description 1
- DSSYKIVIOFKYAU-XCBNKYQSSA-N (R)-camphor Chemical compound C1C[C@@]2(C)C(=O)C[C@@H]1C2(C)C DSSYKIVIOFKYAU-XCBNKYQSSA-N 0.000 description 1
- UHDGCWIWMRVCDJ-UHFFFAOYSA-N 1-beta-D-Xylofuranosyl-NH-Cytosine Natural products O=C1N=C(N)C=CN1C1C(O)C(O)C(CO)O1 UHDGCWIWMRVCDJ-UHFFFAOYSA-N 0.000 description 1
- FPIPGXGPPPQFEQ-UHFFFAOYSA-N 13-cis retinol Natural products OCC=C(C)C=CC=C(C)C=CC1=C(C)CCCC1(C)C FPIPGXGPPPQFEQ-UHFFFAOYSA-N 0.000 description 1
- IHPYMWDTONKSCO-UHFFFAOYSA-N 2,2'-piperazine-1,4-diylbisethanesulfonic acid Chemical compound OS(=O)(=O)CCN1CCN(CCS(O)(=O)=O)CC1 IHPYMWDTONKSCO-UHFFFAOYSA-N 0.000 description 1
- ASJSAQIRZKANQN-CRCLSJGQSA-N 2-deoxy-D-ribose Chemical group OC[C@@H](O)[C@@H](O)CC=O ASJSAQIRZKANQN-CRCLSJGQSA-N 0.000 description 1
- LBLYYCQCTBFVLH-UHFFFAOYSA-M 2-methylbenzenesulfonate Chemical compound CC1=CC=CC=C1S([O-])(=O)=O LBLYYCQCTBFVLH-UHFFFAOYSA-M 0.000 description 1
- 229940080296 2-naphthalenesulfonate Drugs 0.000 description 1
- ZRPLANDPDWYOMZ-UHFFFAOYSA-N 3-cyclopentylpropionic acid Chemical class OC(=O)CCC1CCCC1 ZRPLANDPDWYOMZ-UHFFFAOYSA-N 0.000 description 1
- XMIIGOLPHOKFCH-UHFFFAOYSA-M 3-phenylpropionate Chemical compound [O-]C(=O)CCC1=CC=CC=C1 XMIIGOLPHOKFCH-UHFFFAOYSA-M 0.000 description 1
- 101710169336 5'-deoxyadenosine deaminase Proteins 0.000 description 1
- FHVDTGUDJYJELY-UHFFFAOYSA-N 6-{[2-carboxy-4,5-dihydroxy-6-(phosphanyloxy)oxan-3-yl]oxy}-4,5-dihydroxy-3-phosphanyloxane-2-carboxylic acid Chemical class O1C(C(O)=O)C(P)C(O)C(O)C1OC1C(C(O)=O)OC(OP)C(O)C1O FHVDTGUDJYJELY-UHFFFAOYSA-N 0.000 description 1
- 230000005730 ADP ribosylation Effects 0.000 description 1
- 108020005176 AU Rich Elements Proteins 0.000 description 1
- QTBSBXVTEAMEQO-UHFFFAOYSA-M Acetate Chemical compound CC([O-])=O QTBSBXVTEAMEQO-UHFFFAOYSA-M 0.000 description 1
- 102000007469 Actins Human genes 0.000 description 1
- 108010085238 Actins Proteins 0.000 description 1
- 102100036664 Adenosine deaminase Human genes 0.000 description 1
- GUBGYTABKSRVRQ-XLOQQCSPSA-N Alpha-Lactose Chemical compound O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@H]1O[C@@H]1[C@@H](CO)O[C@H](O)[C@H](O)[C@H]1O GUBGYTABKSRVRQ-XLOQQCSPSA-N 0.000 description 1
- 102100029470 Apolipoprotein E Human genes 0.000 description 1
- 101710095339 Apolipoprotein E Proteins 0.000 description 1
- CIWBSHSKHKDKBQ-JLAZNSOCSA-N Ascorbic acid Natural products OC[C@H](O)[C@H]1OC(=O)C(O)=C1O CIWBSHSKHKDKBQ-JLAZNSOCSA-N 0.000 description 1
- 206010003591 Ataxia Diseases 0.000 description 1
- 208000017785 Autosomal dominant epilepsy with auditory features Diseases 0.000 description 1
- 208000008882 Benign Neonatal Epilepsy Diseases 0.000 description 1
- BTBUEUYNUDRHOZ-UHFFFAOYSA-N Borate Chemical compound [O-]B([O-])[O-] BTBUEUYNUDRHOZ-UHFFFAOYSA-N 0.000 description 1
- 208000014644 Brain disease Diseases 0.000 description 1
- 101100171060 Caenorhabditis elegans div-1 gene Proteins 0.000 description 1
- OYPRJOBELJOOCE-UHFFFAOYSA-N Calcium Chemical compound [Ca] OYPRJOBELJOOCE-UHFFFAOYSA-N 0.000 description 1
- 108090000994 Catalytic RNA Proteins 0.000 description 1
- 102000053642 Catalytic RNA Human genes 0.000 description 1
- 108091005462 Cation channels Proteins 0.000 description 1
- 108091006146 Channels Proteins 0.000 description 1
- 241000723346 Cinnamomum camphora Species 0.000 description 1
- 108091035707 Consensus sequence Proteins 0.000 description 1
- 229920002785 Croscarmellose sodium Polymers 0.000 description 1
- MIKUYHXYGGJMLM-GIMIYPNGSA-N Crotonoside Natural products C1=NC2=C(N)NC(=O)N=C2N1[C@H]1O[C@@H](CO)[C@H](O)[C@@H]1O MIKUYHXYGGJMLM-GIMIYPNGSA-N 0.000 description 1
- UHDGCWIWMRVCDJ-PSQAKQOGSA-N Cytidine Natural products O=C1N=C(N)C=CN1[C@@H]1[C@@H](O)[C@@H](O)[C@H](CO)O1 UHDGCWIWMRVCDJ-PSQAKQOGSA-N 0.000 description 1
- FBPFZTCFMRRESA-FSIIMWSLSA-N D-Glucitol Natural products OC[C@H](O)[C@H](O)[C@@H](O)[C@H](O)CO FBPFZTCFMRRESA-FSIIMWSLSA-N 0.000 description 1
- FBPFZTCFMRRESA-KVTDHHQDSA-N D-Mannitol Chemical compound OC[C@@H](O)[C@@H](O)[C@H](O)[C@H](O)CO FBPFZTCFMRRESA-KVTDHHQDSA-N 0.000 description 1
- ZZZCUOFIHGPKAK-UHFFFAOYSA-N D-erythro-ascorbic acid Natural products OCC1OC(=O)C(O)=C1O ZZZCUOFIHGPKAK-UHFFFAOYSA-N 0.000 description 1
- FBPFZTCFMRRESA-JGWLITMVSA-N D-glucitol Chemical compound OC[C@H](O)[C@@H](O)[C@H](O)[C@H](O)CO FBPFZTCFMRRESA-JGWLITMVSA-N 0.000 description 1
- NYHBQMYGNKIUIF-UHFFFAOYSA-N D-guanosine Natural products C1=2NC(N)=NC(=O)C=2N=CN1C1OC(CO)C(O)C1O NYHBQMYGNKIUIF-UHFFFAOYSA-N 0.000 description 1
- HMFHBZSHGGEWLO-SOOFDHNKSA-N D-ribofuranose Chemical compound OC[C@H]1OC(O)[C@H](O)[C@@H]1O HMFHBZSHGGEWLO-SOOFDHNKSA-N 0.000 description 1
- 230000004543 DNA replication Effects 0.000 description 1
- FEWJPZIEWOKRBE-JCYAYHJZSA-N Dextrotartaric acid Chemical compound OC(=O)[C@H](O)[C@@H](O)C(O)=O FEWJPZIEWOKRBE-JCYAYHJZSA-N 0.000 description 1
- 201000007547 Dravet syndrome Diseases 0.000 description 1
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 1
- 208000032274 Encephalopathy Diseases 0.000 description 1
- 239000001856 Ethyl cellulose Substances 0.000 description 1
- ZZSNKZQZMQGXPY-UHFFFAOYSA-N Ethyl cellulose Chemical compound CCOCC1OC(OC)C(OCC)C(OCC)C1OC1C(O)C(O)C(OC)C(CO)O1 ZZSNKZQZMQGXPY-UHFFFAOYSA-N 0.000 description 1
- 241000206602 Eukaryota Species 0.000 description 1
- 208000033497 Familial temporal lobe epilepsy Diseases 0.000 description 1
- 108090000331 Firefly luciferases Proteins 0.000 description 1
- VZCYOOQTPOCHFL-OWOJBTEDSA-N Fumaric acid Chemical compound OC(=O)\C=C\C(O)=O VZCYOOQTPOCHFL-OWOJBTEDSA-N 0.000 description 1
- 108010010803 Gelatin Proteins 0.000 description 1
- 108010009117 Gluk1 kainate receptor Proteins 0.000 description 1
- 208000034308 Grand mal convulsion Diseases 0.000 description 1
- 241000282412 Homo Species 0.000 description 1
- 101000878605 Homo sapiens Low affinity immunoglobulin epsilon Fc receptor Proteins 0.000 description 1
- VEXZGXHMUGYJMC-UHFFFAOYSA-N Hydrochloric acid Chemical compound Cl VEXZGXHMUGYJMC-UHFFFAOYSA-N 0.000 description 1
- CPELXLSAUQHCOX-UHFFFAOYSA-N Hydrogen bromide Chemical compound Br CPELXLSAUQHCOX-UHFFFAOYSA-N 0.000 description 1
- 229920002153 Hydroxypropyl cellulose Polymers 0.000 description 1
- 206010020751 Hypersensitivity Diseases 0.000 description 1
- 206010021750 Infantile Spasms Diseases 0.000 description 1
- 208000035899 Infantile spasms syndrome Diseases 0.000 description 1
- 229930010555 Inosine Natural products 0.000 description 1
- UGQMRVRMYYASKQ-KQYNXXCUSA-N Inosine Chemical compound O[C@@H]1[C@H](O)[C@@H](CO)O[C@H]1N1C2=NC=NC(O)=C2N=C1 UGQMRVRMYYASKQ-KQYNXXCUSA-N 0.000 description 1
- 108091092195 Intron Proteins 0.000 description 1
- 102000004310 Ion Channels Human genes 0.000 description 1
- 108090000862 Ion Channels Proteins 0.000 description 1
- XUJNEKJLAYXESH-REOHCLBHSA-N L-Cysteine Chemical compound SC[C@H](N)C(O)=O XUJNEKJLAYXESH-REOHCLBHSA-N 0.000 description 1
- CIWBSHSKHKDKBQ-JLAZNSOCSA-M L-ascorbate Chemical compound OC[C@H](O)[C@H]1OC(=O)C(O)=C1[O-] CIWBSHSKHKDKBQ-JLAZNSOCSA-M 0.000 description 1
- FFEARJCKVFRZRR-BYPYZUCNSA-N L-methionine Chemical compound CSCC[C@H](N)C(O)=O FFEARJCKVFRZRR-BYPYZUCNSA-N 0.000 description 1
- JVTAAEKCZFNVCJ-UHFFFAOYSA-M Lactate Chemical compound CC(O)C([O-])=O JVTAAEKCZFNVCJ-UHFFFAOYSA-M 0.000 description 1
- GUBGYTABKSRVRQ-QKKXKWKRSA-N Lactose Natural products OC[C@H]1O[C@@H](O[C@H]2[C@H](O)[C@@H](O)C(O)O[C@@H]2CO)[C@H](O)[C@@H](O)[C@H]1O GUBGYTABKSRVRQ-QKKXKWKRSA-N 0.000 description 1
- WHXSMMKQMYFTQS-UHFFFAOYSA-N Lithium Chemical compound [Li] WHXSMMKQMYFTQS-UHFFFAOYSA-N 0.000 description 1
- 102100038007 Low affinity immunoglobulin epsilon Fc receptor Human genes 0.000 description 1
- FYYHWMGAXLPEAU-UHFFFAOYSA-N Magnesium Chemical compound [Mg] FYYHWMGAXLPEAU-UHFFFAOYSA-N 0.000 description 1
- 208000000676 Malformations of Cortical Development Diseases 0.000 description 1
- OFOBLEOULBTSOW-UHFFFAOYSA-L Malonate Chemical compound [O-]C(=O)CC([O-])=O OFOBLEOULBTSOW-UHFFFAOYSA-L 0.000 description 1
- 229930195725 Mannitol Natural products 0.000 description 1
- 102000016193 Metabotropic glutamate receptors Human genes 0.000 description 1
- 108010010914 Metabotropic glutamate receptors Proteins 0.000 description 1
- AFVFQIVMOAPDHO-UHFFFAOYSA-N Methanesulfonic acid Chemical compound CS(O)(=O)=O AFVFQIVMOAPDHO-UHFFFAOYSA-N 0.000 description 1
- 229920000168 Microcrystalline cellulose Polymers 0.000 description 1
- 229920000881 Modified starch Polymers 0.000 description 1
- 241000699660 Mus musculus Species 0.000 description 1
- 229940127523 NMDA Receptor Antagonists Drugs 0.000 description 1
- 229910002651 NO3 Inorganic materials 0.000 description 1
- 206010028980 Neoplasm Diseases 0.000 description 1
- 208000025966 Neurological disease Diseases 0.000 description 1
- NHNBFGGVMKEFGY-UHFFFAOYSA-N Nitrate Chemical compound [O-][N+]([O-])=O NHNBFGGVMKEFGY-UHFFFAOYSA-N 0.000 description 1
- 101710163270 Nuclease Proteins 0.000 description 1
- 108700026244 Open Reading Frames Proteins 0.000 description 1
- MUBZPKHOEPUJKR-UHFFFAOYSA-N Oxalic acid Chemical compound OC(=O)C(O)=O MUBZPKHOEPUJKR-UHFFFAOYSA-N 0.000 description 1
- 239000007990 PIPES buffer Substances 0.000 description 1
- 229910019142 PO4 Inorganic materials 0.000 description 1
- 208000007542 Paresis Diseases 0.000 description 1
- 208000037158 Partial Epilepsies Diseases 0.000 description 1
- 229920002230 Pectic acid Polymers 0.000 description 1
- 241000425347 Phyla <beetle> Species 0.000 description 1
- 239000002202 Polyethylene glycol Substances 0.000 description 1
- XBDQKXXYIPTUBI-UHFFFAOYSA-M Propionate Chemical compound CCC([O-])=O XBDQKXXYIPTUBI-UHFFFAOYSA-M 0.000 description 1
- 102000008993 Prospero homeobox protein 1 Human genes 0.000 description 1
- 108050000980 Prospero homeobox protein 1 Proteins 0.000 description 1
- 102000009572 RNA Polymerase II Human genes 0.000 description 1
- 108010009460 RNA Polymerase II Proteins 0.000 description 1
- 238000010357 RNA editing Methods 0.000 description 1
- 238000003559 RNA-seq method Methods 0.000 description 1
- 238000011529 RT qPCR Methods 0.000 description 1
- 108020004511 Recombinant DNA Proteins 0.000 description 1
- 102000006382 Ribonucleases Human genes 0.000 description 1
- 108010083644 Ribonucleases Proteins 0.000 description 1
- 108091028664 Ribonucleotide Proteins 0.000 description 1
- PYMYPHUHKUWMLA-LMVFSUKVSA-N Ribose Natural products OC[C@@H](O)[C@@H](O)[C@@H](O)C=O PYMYPHUHKUWMLA-LMVFSUKVSA-N 0.000 description 1
- 206010073677 Severe myoclonic epilepsy of infancy Diseases 0.000 description 1
- 229920001800 Shellac Polymers 0.000 description 1
- 108020004682 Single-Stranded DNA Proteins 0.000 description 1
- 229920002472 Starch Polymers 0.000 description 1
- 235000021355 Stearic acid Nutrition 0.000 description 1
- CZMRCDWAGMRECN-UGDNZRGBSA-N Sucrose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 CZMRCDWAGMRECN-UGDNZRGBSA-N 0.000 description 1
- 229930006000 Sucrose Natural products 0.000 description 1
- QAOWNCQODCNURD-UHFFFAOYSA-L Sulfate Chemical compound [O-]S([O-])(=O)=O QAOWNCQODCNURD-UHFFFAOYSA-L 0.000 description 1
- ZMZDMBWJUHKJPS-UHFFFAOYSA-M Thiocyanate anion Chemical compound [S-]C#N ZMZDMBWJUHKJPS-UHFFFAOYSA-M 0.000 description 1
- 208000006633 Tonic-Clonic Epilepsy Diseases 0.000 description 1
- 108700019146 Transgenes Proteins 0.000 description 1
- 208000026911 Tuberous sclerosis complex Diseases 0.000 description 1
- FPIPGXGPPPQFEQ-BOOMUCAASA-N Vitamin A Natural products OC/C=C(/C)\C=C\C=C(\C)/C=C/C1=C(C)CCCC1(C)C FPIPGXGPPPQFEQ-BOOMUCAASA-N 0.000 description 1
- 229930003268 Vitamin C Natural products 0.000 description 1
- 229930003427 Vitamin E Natural products 0.000 description 1
- 201000006791 West syndrome Diseases 0.000 description 1
- 208000027418 Wounds and injury Diseases 0.000 description 1
- TVXBFESIOXBWNM-UHFFFAOYSA-N Xylitol Natural products OCCC(O)C(O)C(O)CCO TVXBFESIOXBWNM-UHFFFAOYSA-N 0.000 description 1
- 240000008042 Zea mays Species 0.000 description 1
- 235000005824 Zea mays ssp. parviglumis Nutrition 0.000 description 1
- 235000002017 Zea mays subsp mays Nutrition 0.000 description 1
- 108010084455 Zeocin Proteins 0.000 description 1
- 238000002679 ablation Methods 0.000 description 1
- 208000003554 absence epilepsy Diseases 0.000 description 1
- 239000002250 absorbent Substances 0.000 description 1
- 230000002745 absorbent Effects 0.000 description 1
- VJHCJDRQFCCTHL-UHFFFAOYSA-N acetic acid 2,3,4,5,6-pentahydroxyhexanal Chemical compound CC(O)=O.OCC(O)C(O)C(O)C(O)C=O VJHCJDRQFCCTHL-UHFFFAOYSA-N 0.000 description 1
- DPXJVFZANSGRMM-UHFFFAOYSA-N acetic acid;2,3,4,5,6-pentahydroxyhexanal;sodium Chemical compound [Na].CC(O)=O.OCC(O)C(O)C(O)C(O)C=O DPXJVFZANSGRMM-UHFFFAOYSA-N 0.000 description 1
- 230000021736 acetylation Effects 0.000 description 1
- 238000006640 acetylation reaction Methods 0.000 description 1
- 230000002378 acidificating effect Effects 0.000 description 1
- 150000001278 adipic acid derivatives Chemical class 0.000 description 1
- 239000011543 agarose gel Substances 0.000 description 1
- 238000000246 agarose gel electrophoresis Methods 0.000 description 1
- 239000003513 alkali Substances 0.000 description 1
- FPIPGXGPPPQFEQ-OVSJKPMPSA-N all-trans-retinol Chemical compound OC\C=C(/C)\C=C\C=C(/C)\C=C\C1=C(C)CCCC1(C)C FPIPGXGPPPQFEQ-OVSJKPMPSA-N 0.000 description 1
- HMFHBZSHGGEWLO-UHFFFAOYSA-N alpha-D-Furanose-Ribose Natural products OCC1OC(O)C(O)C1O HMFHBZSHGGEWLO-UHFFFAOYSA-N 0.000 description 1
- AEMOLEFTQBMNLQ-BKBMJHBISA-N alpha-D-galacturonic acid Chemical compound O[C@H]1O[C@H](C(O)=O)[C@H](O)[C@H](O)[C@H]1O AEMOLEFTQBMNLQ-BKBMJHBISA-N 0.000 description 1
- AWUCVROLDVIAJX-UHFFFAOYSA-N alpha-glycerophosphate Natural products OCC(O)COP(O)(O)=O AWUCVROLDVIAJX-UHFFFAOYSA-N 0.000 description 1
- 229960000723 ampicillin Drugs 0.000 description 1
- AVKUERGKIZMTKX-NJBDSQKTSA-N ampicillin Chemical compound C1([C@@H](N)C(=O)N[C@H]2[C@H]3SC([C@@H](N3C2=O)C(O)=O)(C)C)=CC=CC=C1 AVKUERGKIZMTKX-NJBDSQKTSA-N 0.000 description 1
- 230000003321 amplification Effects 0.000 description 1
- 210000004727 amygdala Anatomy 0.000 description 1
- 238000010171 animal model Methods 0.000 description 1
- 239000005557 antagonist Substances 0.000 description 1
- 239000003242 anti bacterial agent Substances 0.000 description 1
- 230000000181 anti-adherent effect Effects 0.000 description 1
- 239000003911 antiadherent Substances 0.000 description 1
- 229940088710 antibiotic agent Drugs 0.000 description 1
- 239000003963 antioxidant agent Substances 0.000 description 1
- 235000006708 antioxidants Nutrition 0.000 description 1
- 238000013459 approach Methods 0.000 description 1
- 125000003289 ascorbyl group Chemical class [H]O[C@@]([H])(C([H])([H])O*)[C@@]1([H])OC(=O)C(O*)=C1O* 0.000 description 1
- 210000001130 astrocyte Anatomy 0.000 description 1
- 230000001977 ataxic effect Effects 0.000 description 1
- 239000012752 auxiliary agent Substances 0.000 description 1
- 230000006399 behavior Effects 0.000 description 1
- 230000003542 behavioural effect Effects 0.000 description 1
- 201000008181 benign familial infantile epilepsy Diseases 0.000 description 1
- 201000003452 benign familial neonatal epilepsy Diseases 0.000 description 1
- XMIIGOLPHOKFCH-UHFFFAOYSA-N beta-phenylpropanoic acid Natural products OC(=O)CCC1=CC=CC=C1 XMIIGOLPHOKFCH-UHFFFAOYSA-N 0.000 description 1
- 239000011230 binding agent Substances 0.000 description 1
- 230000004071 biological effect Effects 0.000 description 1
- 230000031018 biological processes and functions Effects 0.000 description 1
- 210000004369 blood Anatomy 0.000 description 1
- 239000008280 blood Substances 0.000 description 1
- 230000037396 body weight Effects 0.000 description 1
- 230000006931 brain damage Effects 0.000 description 1
- 231100000874 brain damage Toxicity 0.000 description 1
- 208000029028 brain injury Diseases 0.000 description 1
- 210000005013 brain tissue Anatomy 0.000 description 1
- 239000011575 calcium Substances 0.000 description 1
- 229910052791 calcium Inorganic materials 0.000 description 1
- 229910000019 calcium carbonate Inorganic materials 0.000 description 1
- FATUQANACHZLRT-KMRXSBRUSA-L calcium glucoheptonate Chemical compound [Ca+2].OC[C@@H](O)[C@@H](O)[C@H](O)[C@@H](O)C(O)C([O-])=O.OC[C@@H](O)[C@@H](O)[C@H](O)[C@@H](O)C(O)C([O-])=O FATUQANACHZLRT-KMRXSBRUSA-L 0.000 description 1
- FUFJGUQYACFECW-UHFFFAOYSA-L calcium hydrogenphosphate Chemical compound [Ca+2].OP([O-])([O-])=O FUFJGUQYACFECW-UHFFFAOYSA-L 0.000 description 1
- 230000010221 calcium permeability Effects 0.000 description 1
- CJZGTCYPCWQAJB-UHFFFAOYSA-L calcium stearate Chemical compound [Ca+2].CCCCCCCCCCCCCCCCCC([O-])=O.CCCCCCCCCCCCCCCCCC([O-])=O CJZGTCYPCWQAJB-UHFFFAOYSA-L 0.000 description 1
- 235000013539 calcium stearate Nutrition 0.000 description 1
- 239000008116 calcium stearate Substances 0.000 description 1
- 229930008380 camphor Natural products 0.000 description 1
- 229960000846 camphor Drugs 0.000 description 1
- 210000000234 capsid Anatomy 0.000 description 1
- 239000002775 capsule Substances 0.000 description 1
- 229910052799 carbon Inorganic materials 0.000 description 1
- 239000001768 carboxy methyl cellulose Substances 0.000 description 1
- 230000032823 cell division Effects 0.000 description 1
- 230000033077 cellular process Effects 0.000 description 1
- 229960005091 chloramphenicol Drugs 0.000 description 1
- WIIZWVCIJKGZOK-RKDXNWHRSA-N chloramphenicol Chemical compound ClC(Cl)C(=O)N[C@H](CO)[C@H](O)C1=CC=C([N+]([O-])=O)C=C1 WIIZWVCIJKGZOK-RKDXNWHRSA-N 0.000 description 1
- 235000015165 citric acid Nutrition 0.000 description 1
- 230000007278 cognition impairment Effects 0.000 description 1
- 239000003086 colorant Substances 0.000 description 1
- 238000007906 compression Methods 0.000 description 1
- 230000006835 compression Effects 0.000 description 1
- 238000004590 computer program Methods 0.000 description 1
- 208000015134 congenital hypothalamic hamartoma syndrome Diseases 0.000 description 1
- 208000013500 continuous spikes and waves during sleep Diseases 0.000 description 1
- 238000011340 continuous therapy Methods 0.000 description 1
- 235000005822 corn Nutrition 0.000 description 1
- 239000006071 cream Substances 0.000 description 1
- 229960005168 croscarmellose Drugs 0.000 description 1
- 229960000913 crospovidone Drugs 0.000 description 1
- 239000001767 crosslinked sodium carboxy methyl cellulose Substances 0.000 description 1
- 238000009109 curative therapy Methods 0.000 description 1
- 235000018417 cysteine Nutrition 0.000 description 1
- XUJNEKJLAYXESH-UHFFFAOYSA-N cysteine Natural products SCC(N)C(O)=O XUJNEKJLAYXESH-UHFFFAOYSA-N 0.000 description 1
- UHDGCWIWMRVCDJ-ZAKLUEHWSA-N cytidine Chemical compound O=C1N=C(N)C=CN1[C@H]1[C@H](O)[C@@H](O)[C@H](CO)O1 UHDGCWIWMRVCDJ-ZAKLUEHWSA-N 0.000 description 1
- 210000000805 cytoplasm Anatomy 0.000 description 1
- 229940104302 cytosine Drugs 0.000 description 1
- 230000003247 decreasing effect Effects 0.000 description 1
- 230000000593 degrading effect Effects 0.000 description 1
- 239000005547 deoxyribonucleotide Substances 0.000 description 1
- 125000002637 deoxyribonucleotide group Chemical group 0.000 description 1
- 238000013461 design Methods 0.000 description 1
- 230000001687 destabilization Effects 0.000 description 1
- 238000001514 detection method Methods 0.000 description 1
- 238000011161 development Methods 0.000 description 1
- 230000018109 developmental process Effects 0.000 description 1
- 238000003745 diagnosis Methods 0.000 description 1
- 239000007884 disintegrant Substances 0.000 description 1
- 239000002270 dispersing agent Substances 0.000 description 1
- POULHZVOKOAJMA-UHFFFAOYSA-M dodecanoate Chemical compound CCCCCCCCCCCC([O-])=O POULHZVOKOAJMA-UHFFFAOYSA-M 0.000 description 1
- 239000008298 dragée Substances 0.000 description 1
- 239000003937 drug carrier Substances 0.000 description 1
- 239000000975 dye Substances 0.000 description 1
- 238000003372 electrophysiological method Methods 0.000 description 1
- 238000004520 electroporation Methods 0.000 description 1
- 239000003974 emollient agent Substances 0.000 description 1
- 239000003995 emulsifying agent Substances 0.000 description 1
- 230000002708 enhancing effect Effects 0.000 description 1
- 201000002933 epilepsy with generalized tonic-clonic seizures Diseases 0.000 description 1
- CCIVGXIOQKPBKL-UHFFFAOYSA-M ethanesulfonate Chemical compound CCS([O-])(=O)=O CCIVGXIOQKPBKL-UHFFFAOYSA-M 0.000 description 1
- 235000019325 ethyl cellulose Nutrition 0.000 description 1
- 229920001249 ethyl cellulose Polymers 0.000 description 1
- 210000003527 eukaryotic cell Anatomy 0.000 description 1
- 230000003492 excitotoxic effect Effects 0.000 description 1
- 231100000063 excitotoxicity Toxicity 0.000 description 1
- 230000001747 exhibiting effect Effects 0.000 description 1
- 238000013401 experimental design Methods 0.000 description 1
- 238000002474 experimental method Methods 0.000 description 1
- 239000013613 expression plasmid Substances 0.000 description 1
- 230000002349 favourable effect Effects 0.000 description 1
- 206010016284 febrile convulsion Diseases 0.000 description 1
- 239000000945 filler Substances 0.000 description 1
- 239000000796 flavoring agent Substances 0.000 description 1
- 235000019634 flavors Nutrition 0.000 description 1
- 238000002073 fluorescence micrograph Methods 0.000 description 1
- 229910052731 fluorine Inorganic materials 0.000 description 1
- 235000003599 food sweetener Nutrition 0.000 description 1
- 238000009472 formulation Methods 0.000 description 1
- 239000003205 fragrance Substances 0.000 description 1
- 210000001652 frontal lobe Anatomy 0.000 description 1
- 229960003692 gamma aminobutyric acid Drugs 0.000 description 1
- BTCSSZJGUNDROE-UHFFFAOYSA-N gamma-aminobutyric acid Chemical compound NCCCC(O)=O BTCSSZJGUNDROE-UHFFFAOYSA-N 0.000 description 1
- WIGCFUFOHFEKBI-UHFFFAOYSA-N gamma-tocopherol Natural products CC(C)CCCC(C)CCCC(C)CCCC1CCC2C(C)C(O)C(C)C(C)C2O1 WIGCFUFOHFEKBI-UHFFFAOYSA-N 0.000 description 1
- 239000008273 gelatin Substances 0.000 description 1
- 229920000159 gelatin Polymers 0.000 description 1
- 239000007903 gelatin capsule Substances 0.000 description 1
- 235000019322 gelatine Nutrition 0.000 description 1
- 235000011852 gelatine desserts Nutrition 0.000 description 1
- 108091008053 gene clusters Proteins 0.000 description 1
- 238000012226 gene silencing method Methods 0.000 description 1
- 230000035784 germination Effects 0.000 description 1
- 210000001362 glutamatergic neuron Anatomy 0.000 description 1
- 230000013595 glycosylation Effects 0.000 description 1
- 238000006206 glycosylation reaction Methods 0.000 description 1
- 229940029575 guanosine Drugs 0.000 description 1
- 201000011066 hemangioma Diseases 0.000 description 1
- 206010019465 hemiparesis Diseases 0.000 description 1
- IPCSVZSSVZVIGE-UHFFFAOYSA-M hexadecanoate Chemical compound CCCCCCCCCCCCCCCC([O-])=O IPCSVZSSVZVIGE-UHFFFAOYSA-M 0.000 description 1
- FUZZWVXGSFPDMH-UHFFFAOYSA-N hexanoic acid Chemical compound CCCCCC(O)=O FUZZWVXGSFPDMH-UHFFFAOYSA-N 0.000 description 1
- 230000036571 hydration Effects 0.000 description 1
- 238000006703 hydration reaction Methods 0.000 description 1
- XMBWDFGMSWQBCA-UHFFFAOYSA-N hydrogen iodide Chemical compound I XMBWDFGMSWQBCA-UHFFFAOYSA-N 0.000 description 1
- ZMZDMBWJUHKJPS-UHFFFAOYSA-N hydrogen thiocyanate Natural products SC#N ZMZDMBWJUHKJPS-UHFFFAOYSA-N 0.000 description 1
- 239000001863 hydroxypropyl cellulose Substances 0.000 description 1
- 235000010977 hydroxypropyl cellulose Nutrition 0.000 description 1
- 239000001866 hydroxypropyl methyl cellulose Substances 0.000 description 1
- 235000010979 hydroxypropyl methyl cellulose Nutrition 0.000 description 1
- 229920003088 hydroxypropyl methyl cellulose Polymers 0.000 description 1
- UFVKGYZPFZQRLF-UHFFFAOYSA-N hydroxypropyl methyl cellulose Chemical compound OC1C(O)C(OC)OC(CO)C1OC1C(O)C(O)C(OC2C(C(O)C(OC3C(C(O)C(O)C(CO)O3)O)C(CO)O2)O)C(CO)O1 UFVKGYZPFZQRLF-UHFFFAOYSA-N 0.000 description 1
- 230000001660 hyperkinetic effect Effects 0.000 description 1
- 238000003384 imaging method Methods 0.000 description 1
- 238000010166 immunofluorescence Methods 0.000 description 1
- 238000010874 in vitro model Methods 0.000 description 1
- 229910052738 indium Inorganic materials 0.000 description 1
- 230000002757 inflammatory effect Effects 0.000 description 1
- 238000001802 infusion Methods 0.000 description 1
- 239000004615 ingredient Substances 0.000 description 1
- 238000011221 initial treatment Methods 0.000 description 1
- 230000000977 initiatory effect Effects 0.000 description 1
- 239000000976 ink Substances 0.000 description 1
- 150000007529 inorganic bases Chemical class 0.000 description 1
- 229960003786 inosine Drugs 0.000 description 1
- 230000003993 interaction Effects 0.000 description 1
- 230000002452 interceptive effect Effects 0.000 description 1
- 238000011835 investigation Methods 0.000 description 1
- 150000002500 ions Chemical class 0.000 description 1
- 230000002427 irreversible effect Effects 0.000 description 1
- 230000007794 irritation Effects 0.000 description 1
- SUMDYPCJJOFFON-UHFFFAOYSA-N isethionic acid Chemical compound OCCS(O)(=O)=O SUMDYPCJJOFFON-UHFFFAOYSA-N 0.000 description 1
- 229930027917 kanamycin Natural products 0.000 description 1
- SBUJHOSQTJFQJX-NOAMYHISSA-N kanamycin Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CN)O[C@@H]1O[C@H]1[C@H](O)[C@@H](O[C@@H]2[C@@H]([C@@H](N)[C@H](O)[C@@H](CO)O2)O)[C@H](N)C[C@@H]1N SBUJHOSQTJFQJX-NOAMYHISSA-N 0.000 description 1
- 229960000318 kanamycin Drugs 0.000 description 1
- 229930182823 kanamycin A Natural products 0.000 description 1
- 229940001447 lactate Drugs 0.000 description 1
- 229940099584 lactobionate Drugs 0.000 description 1
- JYTUSYBCFIZPBE-AMTLMPIISA-N lactobionic acid Chemical compound OC(=O)[C@H](O)[C@@H](O)[C@@H]([C@H](O)CO)O[C@@H]1O[C@H](CO)[C@H](O)[C@H](O)[C@H]1O JYTUSYBCFIZPBE-AMTLMPIISA-N 0.000 description 1
- 239000008101 lactose Substances 0.000 description 1
- 229940070765 laurate Drugs 0.000 description 1
- 239000003446 ligand Substances 0.000 description 1
- 108020001756 ligand binding domains Proteins 0.000 description 1
- 230000000670 limiting effect Effects 0.000 description 1
- 238000001638 lipofection Methods 0.000 description 1
- 229910052744 lithium Inorganic materials 0.000 description 1
- 238000011068 loading method Methods 0.000 description 1
- 230000004807 localization Effects 0.000 description 1
- 239000006210 lotion Substances 0.000 description 1
- 230000016089 mRNA destabilization Effects 0.000 description 1
- 239000011777 magnesium Substances 0.000 description 1
- 229910052749 magnesium Inorganic materials 0.000 description 1
- 235000019359 magnesium stearate Nutrition 0.000 description 1
- 229940049920 malate Drugs 0.000 description 1
- VZCYOOQTPOCHFL-UPHRSURJSA-N maleic acid Chemical compound OC(=O)\C=C/C(O)=O VZCYOOQTPOCHFL-UPHRSURJSA-N 0.000 description 1
- BJEPYKJPYRNKOW-UHFFFAOYSA-N malic acid Chemical compound OC(=O)C(O)CC(O)=O BJEPYKJPYRNKOW-UHFFFAOYSA-N 0.000 description 1
- VQHSOMBJVWLPSR-WUJBLJFYSA-N maltitol Chemical compound OC[C@H](O)[C@@H](O)[C@@H]([C@H](O)CO)O[C@H]1O[C@H](CO)[C@@H](O)[C@H](O)[C@H]1O VQHSOMBJVWLPSR-WUJBLJFYSA-N 0.000 description 1
- 239000000845 maltitol Substances 0.000 description 1
- 235000010449 maltitol Nutrition 0.000 description 1
- 229940035436 maltitol Drugs 0.000 description 1
- 210000004962 mammalian cell Anatomy 0.000 description 1
- 238000007726 management method Methods 0.000 description 1
- 239000000594 mannitol Substances 0.000 description 1
- 235000010355 mannitol Nutrition 0.000 description 1
- 239000003550 marker Substances 0.000 description 1
- 238000002483 medication Methods 0.000 description 1
- 239000012528 membrane Substances 0.000 description 1
- 210000004379 membrane Anatomy 0.000 description 1
- HEBKCHPVOIAQTA-UHFFFAOYSA-N meso ribitol Natural products OCC(O)C(O)C(O)CO HEBKCHPVOIAQTA-UHFFFAOYSA-N 0.000 description 1
- 229930182817 methionine Natural products 0.000 description 1
- 235000006109 methionine Nutrition 0.000 description 1
- 229920000609 methyl cellulose Polymers 0.000 description 1
- 235000010270 methyl p-hydroxybenzoate Nutrition 0.000 description 1
- 239000004292 methyl p-hydroxybenzoate Substances 0.000 description 1
- 239000001923 methylcellulose Substances 0.000 description 1
- 235000010981 methylcellulose Nutrition 0.000 description 1
- 229960002216 methylparaben Drugs 0.000 description 1
- 235000019813 microcrystalline cellulose Nutrition 0.000 description 1
- 239000008108 microcrystalline cellulose Substances 0.000 description 1
- 229940016286 microcrystalline cellulose Drugs 0.000 description 1
- 230000001617 migratory effect Effects 0.000 description 1
- 238000010369 molecular cloning Methods 0.000 description 1
- 125000004573 morpholin-4-yl group Chemical group N1(CCOCC1)* 0.000 description 1
- 230000004660 morphological change Effects 0.000 description 1
- 238000010172 mouse model Methods 0.000 description 1
- 230000007498 myristoylation Effects 0.000 description 1
- KVBGVZZKJNLNJU-UHFFFAOYSA-M naphthalene-2-sulfonate Chemical compound C1=CC=CC2=CC(S(=O)(=O)[O-])=CC=C21 KVBGVZZKJNLNJU-UHFFFAOYSA-M 0.000 description 1
- 208000022145 neurocutaneous syndrome Diseases 0.000 description 1
- BPGXUIVWLQTVLZ-OFGSCBOVSA-N neuropeptide y(npy) Chemical compound C([C@@H](C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)CC)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCCN=C(N)N)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(O)=O)NC(=O)[C@H](CC=1N=CNC=1)NC(=O)[C@H](CCCN=C(N)N)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](C)NC(=O)[C@H](CO)NC(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)[C@H](CC=1C=CC(O)=CC=1)NC(=O)[C@H](CCCN=C(N)N)NC(=O)[C@H](C)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](C)NC(=O)[C@H]1N(CCC1)C(=O)[C@H](C)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)CNC(=O)[C@H]1N(CCC1)C(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H]1N(CCC1)C(=O)[C@H](CCCCN)NC(=O)[C@H](CO)NC(=O)[C@H]1N(CCC1)C(=O)[C@@H](N)CC=1C=CC(O)=CC=1)C1=CC=C(O)C=C1 BPGXUIVWLQTVLZ-OFGSCBOVSA-N 0.000 description 1
- 108091027963 non-coding RNA Proteins 0.000 description 1
- 102000042567 non-coding RNA Human genes 0.000 description 1
- 230000030147 nuclear export Effects 0.000 description 1
- 238000003199 nucleic acid amplification method Methods 0.000 description 1
- 239000002777 nucleoside Substances 0.000 description 1
- OQCDKBAXFALNLD-UHFFFAOYSA-N octadecanoic acid Natural products CCCCCCCC(C)CCCCCCCCC(O)=O OQCDKBAXFALNLD-UHFFFAOYSA-N 0.000 description 1
- 230000009437 off-target effect Effects 0.000 description 1
- 239000002674 ointment Substances 0.000 description 1
- 229940049964 oleate Drugs 0.000 description 1
- ZQPPMHVWECSIRJ-KTKRTIGZSA-N oleic acid Chemical compound CCCCCCCC\C=C/CCCCCCCC(O)=O ZQPPMHVWECSIRJ-KTKRTIGZSA-N 0.000 description 1
- 238000001543 one-way ANOVA Methods 0.000 description 1
- 150000007530 organic bases Chemical class 0.000 description 1
- 210000001769 parahippocampal gyrus Anatomy 0.000 description 1
- 230000001575 pathological effect Effects 0.000 description 1
- 230000009984 peri-natal effect Effects 0.000 description 1
- JRKICGRDRMAZLK-UHFFFAOYSA-L peroxydisulfate Chemical compound [O-]S(=O)(=O)OOS([O-])(=O)=O JRKICGRDRMAZLK-UHFFFAOYSA-L 0.000 description 1
- 239000000825 pharmaceutical preparation Substances 0.000 description 1
- 239000002831 pharmacologic agent Substances 0.000 description 1
- 230000000144 pharmacologic effect Effects 0.000 description 1
- 238000011458 pharmacological treatment Methods 0.000 description 1
- CWCMIVBLVUHDHK-ZSNHEYEWSA-N phleomycin D1 Chemical compound N([C@H](C(=O)N[C@H](C)[C@@H](O)[C@H](C)C(=O)N[C@@H]([C@H](O)C)C(=O)NCCC=1SC[C@@H](N=1)C=1SC=C(N=1)C(=O)NCCCCNC(N)=N)[C@@H](O[C@H]1[C@H]([C@@H](O)[C@H](O)[C@H](CO)O1)O[C@@H]1[C@H]([C@@H](OC(N)=O)[C@H](O)[C@@H](CO)O1)O)C=1N=CNC=1)C(=O)C1=NC([C@H](CC(N)=O)NC[C@H](N)C(N)=O)=NC(N)=C1C CWCMIVBLVUHDHK-ZSNHEYEWSA-N 0.000 description 1
- NBIIXXVUZAFLBC-UHFFFAOYSA-K phosphate Chemical compound [O-]P([O-])([O-])=O NBIIXXVUZAFLBC-UHFFFAOYSA-K 0.000 description 1
- 235000021317 phosphate Nutrition 0.000 description 1
- 239000010452 phosphate Substances 0.000 description 1
- 150000004713 phosphodiesters Chemical group 0.000 description 1
- OJMIONKXNSYLSR-UHFFFAOYSA-N phosphorous acid Chemical group OP(O)O OJMIONKXNSYLSR-UHFFFAOYSA-N 0.000 description 1
- 230000026731 phosphorylation Effects 0.000 description 1
- 238000006366 phosphorylation reaction Methods 0.000 description 1
- 230000004962 physiological condition Effects 0.000 description 1
- 229940075930 picrate Drugs 0.000 description 1
- OXNIZHLAWKMVMX-UHFFFAOYSA-M picrate anion Chemical compound [O-]C1=C([N+]([O-])=O)C=C([N+]([O-])=O)C=C1[N+]([O-])=O OXNIZHLAWKMVMX-UHFFFAOYSA-M 0.000 description 1
- 229950010765 pivalate Drugs 0.000 description 1
- IUGYQRQAERSCNH-UHFFFAOYSA-N pivalic acid Chemical compound CC(C)(C)C(O)=O IUGYQRQAERSCNH-UHFFFAOYSA-N 0.000 description 1
- 239000013600 plasmid vector Substances 0.000 description 1
- 229920001223 polyethylene glycol Polymers 0.000 description 1
- 235000013809 polyvinylpolypyrrolidone Nutrition 0.000 description 1
- 229920000523 polyvinylpolypyrrolidone Polymers 0.000 description 1
- 239000011148 porous material Substances 0.000 description 1
- 230000004481 post-translational protein modification Effects 0.000 description 1
- 229940069328 povidone Drugs 0.000 description 1
- 239000003755 preservative agent Substances 0.000 description 1
- 238000007639 printing Methods 0.000 description 1
- 208000037821 progressive disease Diseases 0.000 description 1
- 210000001236 prokaryotic cell Anatomy 0.000 description 1
- 239000004405 propyl p-hydroxybenzoate Substances 0.000 description 1
- 235000010232 propyl p-hydroxybenzoate Nutrition 0.000 description 1
- 229960003415 propylparaben Drugs 0.000 description 1
- 238000002331 protein detection Methods 0.000 description 1
- 150000003212 purines Chemical class 0.000 description 1
- 150000003230 pyrimidines Chemical class 0.000 description 1
- 125000001453 quaternary ammonium group Chemical group 0.000 description 1
- 201000005070 reflex epilepsy Diseases 0.000 description 1
- 238000000611 regression analysis Methods 0.000 description 1
- 230000014493 regulation of gene expression Effects 0.000 description 1
- 101150066583 rep gene Proteins 0.000 description 1
- 230000003362 replicative effect Effects 0.000 description 1
- 239000011769 retinyl palmitate Substances 0.000 description 1
- 229940108325 retinyl palmitate Drugs 0.000 description 1
- 235000019172 retinyl palmitate Nutrition 0.000 description 1
- 230000001177 retroviral effect Effects 0.000 description 1
- 230000002441 reversible effect Effects 0.000 description 1
- 238000012552 review Methods 0.000 description 1
- 239000002336 ribonucleotide Substances 0.000 description 1
- 125000002652 ribonucleotide group Chemical group 0.000 description 1
- 108091092562 ribozyme Proteins 0.000 description 1
- 239000000523 sample Substances 0.000 description 1
- 238000000926 separation method Methods 0.000 description 1
- ZLGIYFNHBLSMPS-ATJNOEHPSA-N shellac Chemical compound OCCCCCC(O)C(O)CCCCCCCC(O)=O.C1C23[C@H](C(O)=O)CCC2[C@](C)(CO)[C@@H]1C(C(O)=O)=C[C@@H]3O ZLGIYFNHBLSMPS-ATJNOEHPSA-N 0.000 description 1
- 239000004208 shellac Substances 0.000 description 1
- 229940113147 shellac Drugs 0.000 description 1
- 235000013874 shellac Nutrition 0.000 description 1
- 230000011664 signaling Effects 0.000 description 1
- 239000000377 silicon dioxide Substances 0.000 description 1
- 235000012239 silicon dioxide Nutrition 0.000 description 1
- 229960001866 silicon dioxide Drugs 0.000 description 1
- 230000007958 sleep Effects 0.000 description 1
- 150000003384 small molecules Chemical class 0.000 description 1
- AWUCVROLDVIAJX-GSVOUGTGSA-N sn-glycerol 3-phosphate Chemical compound OC[C@@H](O)COP(O)(O)=O AWUCVROLDVIAJX-GSVOUGTGSA-N 0.000 description 1
- 235000019812 sodium carboxymethyl cellulose Nutrition 0.000 description 1
- 229920001027 sodium carboxymethylcellulose Polymers 0.000 description 1
- 239000011780 sodium chloride Substances 0.000 description 1
- 239000001509 sodium citrate Substances 0.000 description 1
- NLJMYIDDQXHKNR-UHFFFAOYSA-K sodium citrate Chemical compound O.O.[Na+].[Na+].[Na+].[O-]C(=O)CC(O)(CC([O-])=O)C([O-])=O NLJMYIDDQXHKNR-UHFFFAOYSA-K 0.000 description 1
- 229960001790 sodium citrate Drugs 0.000 description 1
- 235000011083 sodium citrates Nutrition 0.000 description 1
- 239000008109 sodium starch glycolate Substances 0.000 description 1
- 229920003109 sodium starch glycolate Polymers 0.000 description 1
- 229940079832 sodium starch glycolate Drugs 0.000 description 1
- 239000002904 solvent Substances 0.000 description 1
- 239000000600 sorbitol Substances 0.000 description 1
- 229960002920 sorbitol Drugs 0.000 description 1
- 230000009870 specific binding Effects 0.000 description 1
- 230000006641 stabilisation Effects 0.000 description 1
- 238000011105 stabilization Methods 0.000 description 1
- 239000008107 starch Substances 0.000 description 1
- 229940032147 starch Drugs 0.000 description 1
- 235000019698 starch Nutrition 0.000 description 1
- 239000008117 stearic acid Substances 0.000 description 1
- 229960004274 stearic acid Drugs 0.000 description 1
- 239000008174 sterile solution Substances 0.000 description 1
- 238000007920 subcutaneous administration Methods 0.000 description 1
- KDYFGRWQOYBRFD-UHFFFAOYSA-L succinate(2-) Chemical compound [O-]C(=O)CCC([O-])=O KDYFGRWQOYBRFD-UHFFFAOYSA-L 0.000 description 1
- 239000005720 sucrose Substances 0.000 description 1
- 229960004793 sucrose Drugs 0.000 description 1
- 235000000346 sugar Nutrition 0.000 description 1
- 229910052717 sulfur Inorganic materials 0.000 description 1
- 230000010741 sumoylation Effects 0.000 description 1
- 230000004083 survival effect Effects 0.000 description 1
- 239000000375 suspending agent Substances 0.000 description 1
- 230000002459 sustained effect Effects 0.000 description 1
- 239000003765 sweetening agent Substances 0.000 description 1
- 230000000946 synaptic effect Effects 0.000 description 1
- 238000003786 synthesis reaction Methods 0.000 description 1
- 239000006188 syrup Substances 0.000 description 1
- 235000020357 syrup Nutrition 0.000 description 1
- 239000003826 tablet Substances 0.000 description 1
- 239000000454 talc Substances 0.000 description 1
- 229910052623 talc Inorganic materials 0.000 description 1
- 229940033134 talc Drugs 0.000 description 1
- 235000012222 talc Nutrition 0.000 description 1
- 229940095064 tartrate Drugs 0.000 description 1
- CBXCPBUEXACCNR-UHFFFAOYSA-N tetraethylammonium Chemical compound CC[N+](CC)(CC)CC CBXCPBUEXACCNR-UHFFFAOYSA-N 0.000 description 1
- QEMXHQIAXOOASZ-UHFFFAOYSA-N tetramethylammonium Chemical compound C[N+](C)(C)C QEMXHQIAXOOASZ-UHFFFAOYSA-N 0.000 description 1
- 229940124597 therapeutic agent Drugs 0.000 description 1
- 238000011285 therapeutic regimen Methods 0.000 description 1
- 230000004797 therapeutic response Effects 0.000 description 1
- 239000004408 titanium dioxide Substances 0.000 description 1
- 229960005196 titanium dioxide Drugs 0.000 description 1
- 235000010215 titanium dioxide Nutrition 0.000 description 1
- 238000011200 topical administration Methods 0.000 description 1
- 230000005026 transcription initiation Effects 0.000 description 1
- 230000005030 transcription termination Effects 0.000 description 1
- 230000002103 transcriptional effect Effects 0.000 description 1
- 238000001890 transfection Methods 0.000 description 1
- 238000011820 transgenic animal model Methods 0.000 description 1
- 238000011830 transgenic mouse model Methods 0.000 description 1
- 230000008733 trauma Effects 0.000 description 1
- 229910052721 tungsten Inorganic materials 0.000 description 1
- 238000010798 ubiquitination Methods 0.000 description 1
- 230000034512 ubiquitination Effects 0.000 description 1
- ZDPHROOEEOARMN-UHFFFAOYSA-N undecanoic acid Chemical compound CCCCCCCCCCC(O)=O ZDPHROOEEOARMN-UHFFFAOYSA-N 0.000 description 1
- NQPDZGIKBAWPEJ-UHFFFAOYSA-N valeric acid Chemical class CCCCC(O)=O NQPDZGIKBAWPEJ-UHFFFAOYSA-N 0.000 description 1
- 239000003981 vehicle Substances 0.000 description 1
- 238000012795 verification Methods 0.000 description 1
- 108700026220 vif Genes Proteins 0.000 description 1
- 230000029812 viral genome replication Effects 0.000 description 1
- 235000019155 vitamin A Nutrition 0.000 description 1
- 239000011719 vitamin A Substances 0.000 description 1
- 235000019154 vitamin C Nutrition 0.000 description 1
- 239000011718 vitamin C Substances 0.000 description 1
- 235000019165 vitamin E Nutrition 0.000 description 1
- 229940046009 vitamin E Drugs 0.000 description 1
- 239000011709 vitamin E Substances 0.000 description 1
- 229940045997 vitamin a Drugs 0.000 description 1
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 1
- 235000010447 xylitol Nutrition 0.000 description 1
- 239000000811 xylitol Substances 0.000 description 1
- HEBKCHPVOIAQTA-SCDXWVJYSA-N xylitol Chemical compound OC[C@H](O)[C@@H](O)[C@H](O)CO HEBKCHPVOIAQTA-SCDXWVJYSA-N 0.000 description 1
- 229960002675 xylitol Drugs 0.000 description 1
- 229910052727 yttrium Inorganic materials 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/113—Non-coding nucleic acids modulating the expression of genes, e.g. antisense oligonucleotides; Antisense DNA or RNA; Triplex- forming oligonucleotides; Catalytic nucleic acids, e.g. ribozymes; Nucleic acids used in co-suppression or gene silencing
- C12N15/1138—Non-coding nucleic acids modulating the expression of genes, e.g. antisense oligonucleotides; Antisense DNA or RNA; Triplex- forming oligonucleotides; Catalytic nucleic acids, e.g. ribozymes; Nucleic acids used in co-suppression or gene silencing against receptors or cell surface proteins
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K31/00—Medicinal preparations containing organic active ingredients
- A61K31/70—Carbohydrates; Sugars; Derivatives thereof
- A61K31/7088—Compounds having three or more nucleosides or nucleotides
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P25/00—Drugs for disorders of the nervous system
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/10—Type of nucleic acid
- C12N2310/11—Antisense
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/10—Type of nucleic acid
- C12N2310/14—Type of nucleic acid interfering N.A.
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2310/00—Structure or type of the nucleic acid
- C12N2310/50—Physical structure
- C12N2310/53—Physical structure partially self-complementary or closed
- C12N2310/531—Stem-loop; Hairpin
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2320/00—Applications; Uses
- C12N2320/10—Applications; Uses in screening processes
- C12N2320/11—Applications; Uses in screening processes for the determination of target sites, i.e. of active nucleic acids
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2320/00—Applications; Uses
- C12N2320/30—Special therapeutic applications
- C12N2320/31—Combination therapy
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2330/00—Production
- C12N2330/50—Biochemical production, i.e. in a transformed host cell
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2830/00—Vector systems having a special element relevant for transcription
- C12N2830/50—Vector systems having a special element relevant for transcription regulating RNA stability, not being an intron, e.g. poly A signal
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Engineering & Computer Science (AREA)
- Genetics & Genomics (AREA)
- Biomedical Technology (AREA)
- Chemical & Material Sciences (AREA)
- Molecular Biology (AREA)
- Organic Chemistry (AREA)
- Bioinformatics & Cheminformatics (AREA)
- General Engineering & Computer Science (AREA)
- Biotechnology (AREA)
- Wood Science & Technology (AREA)
- Zoology (AREA)
- General Health & Medical Sciences (AREA)
- Plant Pathology (AREA)
- Microbiology (AREA)
- Biophysics (AREA)
- Biochemistry (AREA)
- Physics & Mathematics (AREA)
- Animal Behavior & Ethology (AREA)
- Veterinary Medicine (AREA)
- Pharmacology & Pharmacy (AREA)
- Medicinal Chemistry (AREA)
- Public Health (AREA)
- General Chemical & Material Sciences (AREA)
- Neurology (AREA)
- Neurosurgery (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Epidemiology (AREA)
- Nuclear Medicine, Radiotherapy & Molecular Imaging (AREA)
- Pharmaceuticals Containing Other Organic And Inorganic Compounds (AREA)
- Medicines That Contain Protein Lipid Enzymes And Other Medicines (AREA)
- Saccharide Compounds (AREA)
- Medicines Containing Material From Animals Or Micro-Organisms (AREA)
- Acyclic And Carbocyclic Compounds In Medicinal Compositions (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
Abstract
본원 발명은 Grik2 mRNA를 표적으로 함으로써, 치료가 필요한 개체에서 뇌전증, 예컨대 측두엽 뇌전증을 치료하기 위한 유전자 요법에 관련된 방법과 조성물을 제공한다. 특히, 본원 발명은 GluK2 단백질의 발현을 저해할 수 있고 과흥분 뉴런 회로에서 뇌전증모양 방전을 저해할 수 있는 저해성 RNA 작제물을 제공한다.
Description
서열 목록
본 출원은 서열 목록을 내포하는데, 이것은 ASCII 형식으로 전자적으로 제출되었고 본원에서 온전히 참조로서 편입된다. 2021년 7월 8일 자에 작성된 상기 ASCII 사본은 "51460-003WO4_Sequence_Listing_7_8_21_ST25"로 명명되고 크기에서 425,553 바이트이다.
발명의 분야
본원 발명은 뇌전증의 분야이다. 특히, 본원 발명은 뇌전증, 예컨대 예를 들면, 측두엽 뇌전증을 치료하기 위한 방법과 조성물에 관계한다.
배경
측두엽 뇌전증 (TLE)은 성인에서 부분적인 뇌전증의 가장 흔한 형태 (뇌전증의 모든 형태 중 30-40%)이다. 해마가 TLE의 병태생리에서 핵심적인 역할을 수행하는 것으로 충분히 확립되었다. TLE의 인간 환자 및 동물 모형에서, 뉴런 회로의 일탈적 재배선이 발생한다. 네트워크 재편성 ("반응성 가소성")의 최고 실례 중에서 하나는 재발성 흥분성 루프를 야기하는, 해마 내에 치상 과립 세포 (DGC) 위에 신규한 병리생리학적 글루타민산성 시냅스를 확립하는 재발성 이끼 섬유 (rMF)의 발아이다 (Tauck and Nadler, 1985; Represa et al., 1989a, 1989b; Sutula et al., 1989; Gabriel et al., 2004). rMF 시냅스는 이소성 카이네이트 수용체 (KAR)를 통해 작동한다 (Epsztein et al., 2005; Artinian et al., 2011, 2015). KAR은 GluK1-GluK5 아단위로부터 조립된 사합체성 글루타민산염 수용체이다. 이종성 발현 시스템에서, GluK1, GluK2 및 GluK3은 동종 수용체를 형성할 수 있고, 반면 GluK4 및 GluK5는 GluK1-3 아단위와 함께 이종 수용체를 형성한다. 선천적 KAR은 뇌에서 폭넓게 분포되는데, 높은 밀도의 수용체가 TLE에 관련된 핵심 구조인 해마에서 발견된다 (Carta et al, 2016, EJN). 본 발명자들에 의한 선행 연구는 발작간 극파 및 발작성 방전을 포함하는 뇌전증 활성이, GluK2 KAR 아단위를 결여하는 생쥐에서 유의미하게 감소된다는 것을 확립하였다. 게다가, 이소성 시냅스 KAR을 차단하는, GluK2/GluK5 내포 KAR의 약리학적 소형 분자 길항제의 이용 이후에, 뇌전증모양 활성이 강하게 감소되었다 (Peret et al., 2014). 이들 데이터는 DGC 내에 rMF에서 이소성으로 발현된 KAR이 TLE에서 만성 발작에서 주요한 역할을 수행한다는 것을 증명한다. 이런 이유로, DGC에서 발현되고 GluK2/GluK5로 구성되는 일탈적 KAR은 약제 내성 TLE의 치료를 위한 유망한 표적을 대표하는 것으로 고려된다.
많은 질환 표적에 대해 RNA 간섭 (RNAi) 전략이 제안되었다. RNAi 기반 요법의 성공적인 적용은 제한되었다. RNAi 치료제는 특히, 중추신경계 (CNS)에서 흔히 그러하듯, 복잡한 유전자 발현 패턴이 존재하는 경우에, 복수의 과제 예컨대 RNA 설계를 위한 정보를 알아내기 위한 감수성 표적 이탈 도메인의 예측, 가변적인 생체내 유전자 침묵 효능, 그리고 표적 이탈 효과의 감소에 직면한다. 하지만, 난치성 TLE의 치료에 가용한 RNAi 기반 유전자 요법은 제한된다. 이런 이유로, 발작 장애, 예컨대 예를 들면, TLE (예를 들면, 치료에 불응성인 TLE)의 치료를 위한 새로운 치료적 양상이 시급히 요구된다.
발명의 요약
본원 발명은 치료가 필요한 개체 (예를 들면, 인간)에서 뇌전증, 예컨대 예를 들면, 측두엽 뇌전증 (TLE)의 치료 또는 예방을 위한 조성물과 방법을 제공한다. 개시된 방법은 글루타민산염 이온성 수용체 카이네이트 유형 아단위 2 (Grik2) 유전자에 의해 인코딩된 mRNA를 표적으로 하는 저해성 RNA (예를 들면, 안티센스 올리고뉴클레오티드 (ASO, shRNA, siRNA, 마이크로RNA, 또는 shmiRNA), 또는 이것을 인코딩하는 핵산 벡터 (예를 들면, 렌티바이러스 벡터 또는 아데노 관련 바이러스 (AAV) 벡터, 예컨대 예를 들면, AAV9 벡터)의 치료 효과량을, 뇌전증을 겪는 것으로 진단되거나 또는 뇌전증이 발달할 위험에 처해 있는 개체에게 투여하는 단계를 포함한다. 본원 발명은 또한, 개시된 ASO 작용제 또는 이들을 인코딩하는 핵산 벡터 중에서 하나 이상을 내포하는 약학적 조성물을 특징으로 한다.
첫 번째 양상에서, 본원 발명은 Grik2 mRNA의 단일 가닥 영역 내에 특이적으로 혼성화하는, 800개 이내 (예를 들면, 800, 750, 700, 650, 600, 550, 500, 450, 400, 350, 300, 250, 200, 150, 100, 90, 80, 70, 60, 50, 40, 30, 29, 28, 27, 26, 25, 24, 23, 22, 21, 20, 또는 19개 이내) 뉴클레오티드의 길이를 갖는 단리된 폴리뉴클레오티드를 제공하는데, 여기서 상기 혼성화된 폴리뉴클레오티드는 18 kcal/mol보다 작은 (예를 들면, 17 kcal/mol, 16 kcal/mol, 15 kcal/mol, 14 kcal/mol, 13 kcal/mol, 12 kcal/mol, 11 kcal/mol, 10 kcal/mol, 9 kcal/mol, 8 kcal/mol, 7 kcal/mol, 6 kcal/mol, 5 kcal/mol, 4 kcal/mol, 3 kcal/mol, 2 kcal/mol, 또는 1 kcal/mol 또는 그 미만보다 작은) 표적 개방 에너지를 갖고, 그리고 여기서: (a) 상기 폴리뉴클레오티드는 서열 번호: 772-774 (다시 말하면, 유럽 특허 출원 번호: EP19185533.7의 서열 번호: 1-3) 중에서 하나의 핵산 서열을 포함하지 않거나; (b) 상기 폴리뉴클레오티드는 서열 번호: 68 또는 서열 번호: 68 및 서열 번호: 649의 핵산 서열을 포함하거나; 또는 (c) 상기 폴리뉴클레오티드는 5.53 kcal/mol 및 5.55 kcal/mol 사이에 있는 총 개방 에너지 (예를 들면, 5.4 kcal/mol)를 갖지 않는다.
앞서 말한 양상의 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 1-771의 서열 중에서 하나와 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 서열 번호: 772-774 중에서 하나의 핵산 서열은 5.53 kcal/mol 및 5.55 kcal/mol 사이에 있는 총 개방 에너지 (예를 들면, 5.4 kcal/mol)를 갖는다.
다른 양상에서, 본원 발명은 Grik2 mRNA의 단일 가닥 영역 내에 특이적으로 혼성화하는 23개 이내 뉴클레오티드의 길이를 갖는 단리된 RNA 폴리뉴클레오티드를 제공하는데, 여기서 상기 혼성화된 폴리뉴클레오티드는 18 kcal/mol보다 작은 (예를 들면, 17 kcal/mol, 16 kcal/mol, 15 kcal/mol, 14 kcal/mol, 13 kcal/mol, 12 kcal/mol, 11 kcal/mol, 10 kcal/mol, 9 kcal/mol, 8 kcal/mol, 7 kcal/mol, 6 kcal/mol, 5 kcal/mol, 4 kcal/mol, 3 kcal/mol, 2 kcal/mol, 또는 1 kcal/mol 또는 그 미만보다 작은) 총 개방 에너지를 갖고, 여기서 상기 폴리뉴클레오티드는 서열 번호: 772-774 중에서 하나의 핵산 서열을 포함하지 않는다.
앞서 말한 양상의 일부 구체예에서, 혼성화된 폴리뉴클레오티드는 5.53 kcal/mol 및 5.55 kcal/mol 사이에 있는 총 개방 에너지를 갖지 않는다. 일부 구체예에서, 혼성화된 폴리뉴클레오티드는 5.54 kcal/mol보다 작은 총 개방 에너지를 갖는다. 일부 구체예에서, 혼성화된 폴리뉴클레오티드는 5.54 kcal/mol보다 큰 총 개방 에너지를 갖는다. 일부 구체예에서, 혼성화된 폴리뉴클레오티드는 5.54 kcal/mol보다 작거나 또는 5.54 kcal/mol보다 큰 총 개방 에너지를 갖는다.
앞서 말한 양상의 일부 구체예에서, 혼성화된 폴리뉴클레오티드는 -35 kcal/mol보다 큰 (예를 들면, -30 kcal/mol, -25 kcal/mol, -20 kcal/mol, -15 kcal/mol, -10 kcal/mol, -5 kcal/mol, -4 kcal/mol, -3 kcal/mol, -2 kcal/mol, -1 kcal/mol 또는 그 초과보다 큰) 이중나선 형성의/으로부터 에너지를 갖는다. 일부 구체예에서, 혼성화된 폴리뉴클레오티드는 -36.7 kcal/mol 및 -36.5 kcal/mol 사이에 있는 이중나선 형성의 에너지를 갖지 않는다. 일부 구체예에서, 혼성화된 폴리뉴클레오티드는 -36.61 kcal/mol보다 큰 이중나선 형성의 에너지를 갖는다. 일부 구체예에서, 혼성화된 폴리뉴클레오티드는 -36.61 kcal/mol보다 작은 이중나선 형성의 에너지를 갖는다.
일부 구체예에서, 혼성화된 폴리뉴클레오티드는 -24 kcal/mol보다 큰 (예를 들면, -20 kcal/mol, -15 kcal/mol, -10 kcal/mol, -5 kcal/mol, -4 kcal/mol, 3 kcal/mol, -2 kcal/mol, -1 kcal/mol 또는 그 초과보다 큰) 총 결합 에너지를 갖는다. 일부 구체예에서, 혼성화된 폴리뉴클레오티드는 -29.5 kcal/mol 및 -29.3 kcal/mol 사이에 있는 총 결합 에너지를 갖지 않는다. 일부 구체예에서, 혼성화된 폴리뉴클레오티드는 -29.4 kcal/mol보다 큰 총 결합 에너지를 갖는다. 일부 구체예에서, 혼성화된 폴리뉴클레오티드는 -29.4 kcal/mol보다 작은 총 결합 에너지를 갖는다.
일부 구체예에서, 혼성화된 폴리뉴클레오티드는 50%보다 적은 (예를 들면, 45%, 40%, 35%, 30%, 25%, 20%, 15%, 10%, 5%, 4%, 3%, 2%, 1%, 또는 그 미만보다 적은) GC 함량을 갖는다. 일부 구체예에서, 혼성화된 폴리뉴클레오티드는 42.7% 및 47.6% 사이에 있는 GC 함량을 갖지 않는다. 일부 구체예에서, 혼성화된 폴리뉴클레오티드는 42.9%보다 적은 GC 함량을 갖는다. 일부 구체예에서, 혼성화된 폴리뉴클레오티드는 42.9%보다 많은 GC 함량을 갖는다. 일부 구체예에서 폴리뉴클레오티드에 대한 GC 함량이 결정된다. 일부 구체예에서, 폴리뉴클레오티드에 실제적으로 상보적인 (예를 들면, 10, 9, 8, 7, 6, 5, 4, 3, 2, 또는 1개 이내의 부정합을 갖는) 서열에 대한 GC 함량이 결정된다. 일부 구체예에서, 폴리뉴클레오티드 및 상기 폴리뉴클레오티드에 실제적으로 상보적인 서열 사이의 혼성화에 의해 형성된 이중나선에 대해 GC 함량이 결정된다.
일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 1-771의 서열 중에서 하나 이상과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 68의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 68 및 649의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다.
다른 양상에서, 본원 발명은 Grik2 mRNA의 단일 가닥 영역 내에 특이적으로 혼성화하는 800개 이내 뉴클레오티드의 길이를 갖는 단리된 폴리뉴클레오티드를 제공하는데, 여기서 상기 혼성화된 폴리뉴클레오티드는 5.53 및 5.55 kcal/mol 사이에 있는 총 개방 에너지를 갖지 않고, 그리고 여기서 상기 폴리뉴클레오티드는 서열 번호: 772-774 중에서 하나의 핵산 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 1-771의 서열 중에서 하나 이상과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 68의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 68 및 649의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다.
다른 양상에서, 본원 발명은 Grik2 mRNA의 단일 가닥 영역 내에 특이적으로 혼성화하는 800개 이내 뉴클레오티드의 길이를 갖는 단리된 폴리뉴클레오티드를 제공하는데, 여기서 상기 혼성화된 폴리뉴클레오티드는 -36.7 및 -36.5 kcal/mol 사이에 있는 이중나선 형성의 에너지를 갖지 않고, 그리고 여기서 상기 폴리뉴클레오티드는 서열 번호: 772-774 중에서 하나의 핵산 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 1-771의 서열 중에서 하나 이상과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 68의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 68 및 649의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다.
다른 양상에서, 본원 발명은 Grik2 mRNA의 단일 가닥 영역 내에 특이적으로 혼성화하는 800개 이내 뉴클레오티드의 길이를 갖는 단리된 폴리뉴클레오티드를 제공하는데, 여기서 상기 혼성화된 폴리뉴클레오티드는 -29.5 및 -29.3 kcal/mol 사이에 있는 총 결합 에너지를 갖지 않고, 그리고 여기서 상기 폴리뉴클레오티드는 서열 번호: 772-774 중에서 하나의 핵산 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 1-771의 서열 중에서 하나 이상과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 68의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 68 및 649의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다.
다른 양상에서, 본원 발명은 Grik2 mRNA의 단일 가닥 영역 내에 특이적으로 혼성화하는 23개 이내 뉴클레오티드의 길이를 갖는 단리된 RNA 폴리뉴클레오티드를 제공하는데, 여기서 상기 혼성화된 폴리뉴클레오티드는 5.53 및 5.55 kcal/mol 사이에 있는 총 개방 에너지를 갖지 않고, 그리고 여기서 상기 폴리뉴클레오티드는 서열 번호: 772-774 중에서 하나의 핵산 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 1-771의 서열 중에서 하나 이상과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 68의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 68 및 649의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다.
다른 양상에서, 본원 발명은 Grik2 mRNA의 단일 가닥 영역 내에 특이적으로 혼성화하는 23개 이내 뉴클레오티드의 길이를 갖는 단리된 RNA 폴리뉴클레오티드를 제공하는데, 여기서 상기 혼성화된 폴리뉴클레오티드는 -36.7 및 -36.5 kcal/mol 사이에 있는 이중나선 형성의 에너지를 갖지 않고, 그리고 여기서 상기 폴리뉴클레오티드는 서열 번호: 772-774 중에서 하나의 핵산 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 1-771의 서열 중에서 하나 이상과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 68의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 68 및 649의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다.
다른 양상에서, 본원 발명은 Grik2 mRNA의 단일 가닥 영역 내에 특이적으로 혼성화하는 23개 이내 뉴클레오티드의 길이를 갖는 단리된 RNA 폴리뉴클레오티드를 제공하는데, 여기서 상기 혼성화된 폴리뉴클레오티드는 -29.5 및 -29.3 kcal/mol 사이에 있는 총 결합 에너지를 갖지 않고, 그리고 여기서 상기 폴리뉴클레오티드는 서열 번호: 772-774 중에서 하나의 핵산 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 1-771의 서열 중에서 하나 이상과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 68의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 68 및 649의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다.
앞서 말한 양상의 일부 구체예에서, Grik2 mRNA의 단일 가닥 영역은 루프 영역 1-14로 구성된 군에서 선택된다. 일부 구체예에서, 폴리뉴클레오티드는 (a) Grik2 mRNA의 루프 1 영역; (b) Grik2 mRNA의 루프 2 영역; (c) Grik2 mRNA의 루프 3 영역; (d) Grik2 mRNA의 루프 4 영역; (e) Grik2 mRNA의 루프 5 영역; (f) Grik2 mRNA의 루프 6 영역; (g) Grik2 mRNA의 루프 7 영역; (h) Grik2 mRNA의 루프 8 영역; (i) Grik2 mRNA의 루프 9 영역; (j) Grik2 mRNA의 루프 10 영역; (k) Grik2 mRNA의 루프 11 영역; (l) Grik2 mRNA의 루프 12 영역; (m) Grik2 mRNA의 루프 13 영역; 또는 (n) Grik2 mRNA의 루프 14 영역 내에 특이적으로 혼성화한다.
일부 구체예에서, 루프 1 영역은 서열 번호: 145의 적어도 10개 (예를 들면, 적어도 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩된다.
일부 구체예에서, 루프 2 영역은 서열 번호: 146의 적어도 10개 (예를 들면, 적어도 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩된다.
일부 구체예에서, 루프 3 영역은 서열 번호: 147의 적어도 10개 (예를 들면, 적어도 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩된다.
일부 구체예에서, 루프 4 영역은 서열 번호: 148의 적어도 10개 (예를 들면, 적어도 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩된다.
일부 구체예에서, 루프 5 영역은 서열 번호: 149의 적어도 10개 (예를 들면, 적어도 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩된다.
일부 구체예에서, 루프 6 영역은 서열 번호: 150의 적어도 10개 (예를 들면, 적어도 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩된다.
일부 구체예에서, 루프 7 영역은 서열 번호: 151의 적어도 10개 (예를 들면, 적어도 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩된다.
일부 구체예에서, 루프 8 영역은 서열 번호: 152의 적어도 10개 (예를 들면, 적어도 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩된다.
일부 구체예에서, 루프 9 영역은 서열 번호: 153의 적어도 10개 (예를 들면, 적어도 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩된다.
일부 구체예에서, 루프 10 영역은 서열 번호: 154의 적어도 10개 (예를 들면, 적어도 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩된다.
일부 구체예에서, 루프 11 영역은 서열 번호: 155의 적어도 10개 (예를 들면, 적어도 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩된다.
일부 구체예에서, 루프 12 영역은 서열 번호: 156의 적어도 10개 (예를 들면, 적어도 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩된다.
일부 구체예에서, 루프 13 영역은 서열 번호: 157의 적어도 10개 (예를 들면, 적어도 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩된다.
일부 구체예에서, 루프 14 영역은 서열 번호: 158의 적어도 10개 (예를 들면, 적어도 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩된다.
일부 구체예에서, 서열 동일성은 서열 번호: 145-158 중에서 하나의 적어도 15개 (예를 들면, 적어도 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 결정된다. 일부 구체예에서, 서열 동일성은 서열 번호: 145-158 중에서 하나의 적어도 30개 (예를 들면, 적어도 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 결정된다. 일부 구체예에서, 서열 동일성은 서열 번호: 145-158 중에서 하나의 적어도 60개 (예를 들면, 적어도 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 결정된다. 일부 구체예에서, 서열 동일성은 서열 번호: 145-158 중에서 하나의 전장에 걸쳐 결정된다.
일부 구체예에서, 폴리뉴클레오티드는 다음을 포함한다:
(a)
서열 번호: 1의 적어도 5개 (예를 들면, 적어도 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(b)
서열 번호: 4의 적어도 5개 (예를 들면, 적어도 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(c)
(i) 서열 번호: 5의 적어도 5개 (예를 들면, 적어도 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 또는
(ii)서열 번호: 6의 적어도 5개 (예를 들면, 적어도 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(d)
서열 번호: 7의 적어도 5개 (예를 들면, 적어도 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(e)
서열 번호: 96의 적어도 5개 (예를 들면, 적어도 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(f)
(i) 서열 번호: 8의 적어도 5개 (예를 들면, 적어도 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 98의 적어도 5개 (예를 들면, 적어도 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 또는
(iii) 서열 번호: 99의 적어도 5개 (예를 들면, 적어도 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(g)
서열 번호: 9의 적어도 5개 (예를 들면, 적어도 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(h)
서열 번호: 63의 적어도 5개 (예를 들면, 적어도 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(i)
서열 번호: 10의 적어도 5개 (예를 들면, 적어도 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 또는
(j)
서열 번호: 11의 적어도 5개 (예를 들면, 적어도 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열.
일부 구체예에서, 서열 동일성은 서열 번호: 1, 411, 63, 96, 98 또는 99 중에서 하나의 적어도 10개 (예를 들면, 적어도 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 결정된다. 일부 구체예에서, 서열 동일성은 서열 번호: 1, 4-11, 63, 96, 98 또는 99 중에서 하나의 적어도 15개 (예를 들면, 적어도 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 결정된다. 일부 구체예에서, 서열 동일성은 서열 번호: 1, 4-11, 63, 96, 98 또는 99 중에서 하나의 적어도 20개 (예를 들면, 적어도 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 결정된다. 일부 구체예에서, 서열 동일성은 서열 번호: 1, 4-11, 63, 96, 98 또는 99 중에서 하나의 전장에 걸쳐 결정된다.
일부 구체예에서, 폴리뉴클레오티드는 상기 폴리뉴클레오티드 및 Grik2 mRNA의 단일 가닥 영역에 의해 형성된 이중나선 구조를 포함하고, 여기서 상기 이중나선 구조는 상기 폴리뉴클레오티드의 뉴클레오티드 및 Grik2 mRNA의 단일 가닥 영역의 뉴클레오티드 사이에 적어도 하나 (예를 들면, 적어도 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15개 또는 그 초과)의 부정합을 포함한다.
일부 구체예에서, Grik2 mRNA의 단일 가닥 영역은 루프 영역 1-14로 구성된 군에서 선택된다.
일부 구체예에서, 평균 위치 엔트로피는 23개 내지 79개 뉴클레오티드에 걸쳐 계산된다.
일부 구체예에서, Grik2 mRNA의 단일 가닥 영역은 비대합 영역 1-5로 구성된 군에서 선택된다.
일부 구체예에서, 폴리뉴클레오티드는 (a) Grik2 mRNA의 비대합 영역 1; (b) Grik2 mRNA의 비대합 영역 2; (c) Grik2 mRNA의 비대합 영역 3; (d) Grik2 mRNA의 비대합 영역 4; 또는 (e) Grik2 mRNA의 비대합 영역 5 내에 특이적으로 혼성화한다.
일부 구체예에서, (a) 비대합 영역 1은 서열 번호: 159의 적어도 10개 (예를 들면, 적어도 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고; (b) 비대합 영역 2는 서열 번호: 160의 적어도 10개 (예를 들면, 적어도 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고; (c) 비대합 영역 3은 서열 번호: 161의 적어도 10개 (예를 들면, 적어도 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고; (d) 비대합 영역 4는 서열 번호: 162의 적어도 10개 (예를 들면, 적어도 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고; 및/또는 (e) 비대합 영역 5는 서열 번호: 163의 적어도 10개 (예를 들면, 적어도 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩된다.
일부 구체예에서, 서열 동일성은 서열 번호: 159-163 중에서 하나의 적어도 15개 (예를 들면, 적어도 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 결정된다. 일부 구체예에서, 서열 동일성은 서열 번호: 159-163 중에서 하나의 적어도 30개 (예를 들면, 적어도 35, 40, 45, 50, 55, 60, 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 결정된다. 일부 구체예에서, 서열 동일성은 서열 번호: 159-163 중에서 하나의 적어도 60개 (예를 들면, 적어도 65, 70, 75, 또는 80개)의 연속 뉴클레오티드에 걸쳐 결정된다. 일부 구체예에서, 서열 동일성은 서열 번호: 159-163 중에서 하나의 전장에 걸쳐 결정된다.
일부 구체예에서, 폴리뉴클레오티드는 다음을 포함한다:
(a)
(i) 서열 번호: 13의 적어도 5개 (예를 들면, 적어도 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 14의 적어도 5개 (예를 들면, 적어도 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iii) 서열 번호: 72의 적어도 5개 (예를 들면, 적어도 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 또는
(iv) 서열 번호: 73의 적어도 5개 (예를 들면, 적어도 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(b)
서열 번호: 15의 적어도 5개 (예를 들면, 적어도 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 또는
(c)
서열 번호: 16의 적어도 5개 (예를 들면, 적어도 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열.
일부 구체예에서, 서열 동일성은 서열 번호: 13-16, 72 또는 73 중에서 하나의 적어도 10개 (예를 들면, 적어도 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 결정된다. 일부 구체예에서, 서열 동일성은 서열 번호: 13-16, 72 또는 73 중에서 하나의 적어도 15개 (예를 들면, 적어도 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 결정된다. 일부 구체예에서, 서열 동일성은 서열 번호: 13-16, 72 또는 73 중에서 하나의 적어도 20개 (예를 들면, 적어도 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 결정된다. 일부 구체예에서, 서열 동일성은 서열 번호: 13-16, 72 또는 73 중에서 하나의 전장에 걸쳐 결정된다. 일부 구체예에서, 서열 동일성은 서열 번호: 13-16, 72, 또는 73 중에서 하나의 30개 이내 (예를 들면, 30, 25, 20, 15, 10, 9, 8, 7, 6, 5, 4, 3, 또는 2개 이내)의 연속 뉴클레오티드에 걸쳐 결정된다. 일부 구체예에서, 서열 동일성은 서열 번호: 13-16, 72, 또는 73 중에서 하나의 25개 이내 (예를 들면, 25, 20, 15, 10, 9, 8, 7, 6, 5, 4, 3, 또는 2개 이내)의 연속 뉴클레오티드에 걸쳐 결정된다.
일부 구체예에서, 폴리뉴클레오티드는 상기 폴리뉴클레오티드 및 Grik2 mRNA의 단일 가닥 영역에 의해 형성된 이중나선 구조를 포함하고, 여기서 상기 이중나선 구조는 상기 폴리뉴클레오티드의 뉴클레오티드 및 Grik2 mRNA의 단일 가닥 영역의 뉴클레오티드 사이에 적어도 하나 (예를 들면, 적어도 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15개 또는 그 초과)의 부정합을 포함한다.
일부 구체예에서, 평균 위치 엔트로피는 23개 내지 79개 뉴클레오티드에 걸쳐 계산된다.
일부 구체예에서, 폴리뉴클레오티드는 Grik2 mRNA의 코딩 서열에 혼성화한다. 일부 구체예에서, 폴리뉴클레오티드는 (a) Grik2 mRNA의 엑손 1 내에 영역; (b) Grik2 mRNA의 엑손 2 내에 영역; (c) Grik2 mRNA의 엑손 3 내에 영역; (d) Grik2 mRNA의 엑손 4 내에 영역; (e) Grik2 mRNA의 엑손 5 내에 영역; (f) Grik2 mRNA의 엑손 6 내에 영역; (g) Grik2 mRNA의 엑손 7 내에 영역; (h) Grik2 mRNA의 엑손 8 내에 영역; (i) Grik2 mRNA의 엑손 9 내에 영역; (j) Grik2 mRNA의 엑손 10 내에 영역; (k) Grik2 mRNA의 엑손 11 내에 영역; (l) Grik2 mRNA의 엑손 12 내에 영역; (m) Grik2 mRNA의 엑손 13 내에 영역; (n) Grik2 mRNA의 엑손 14 내에 영역; (o) Grik2 mRNA의 엑손 15 내에 영역; 및/또는 (p) Grik2 mRNA의 엑손 16 내에 영역에 혼성화한다.
일부 구체예에서, (a) Grik2 mRNA의 엑손 1 서열 번호: 129의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고; (b) Grik2 mRNA의 엑손 2는 서열 번호: 130의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고; (c) Grik2 mRNA의 엑손 3은 서열 번호: 131의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고; (d) Grik2 mRNA의 엑손 4는 서열 번호: 132의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고; (e) Grik2 mRNA의 엑손 5는 서열 번호: 133의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고; (f) Grik2 mRNA의 엑손 6은 서열 번호: 134의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고; (g) Grik2 mRNA의 엑손 7은 서열 번호: 135의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고; (h) Grik2 mRNA의 엑손 8은 서열 번호: 136의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고; (i) Grik2 mRNA의 엑손 9는 서열 번호: 137의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고; (j) Grik2 mRNA의 엑손 10은 서열 번호: 138의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고; (k) Grik2 mRNA의 엑손 11은 서열 번호: 139의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고; (l) Grik2 mRNA의 엑손 12는 서열 번호: 140의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고; (m) Grik2 mRNA의 엑손 13은 서열 번호: 141의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고; (n) Grik2 mRNA의 엑손 14는 서열 번호: 142의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고; (o) Grik2 mRNA의 엑손 15는 서열 번호: 143의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고; 및/또는 (p) Grik2 mRNA의 엑손 16은 서열 번호: 144의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩된다.
일부 구체예에서, 폴리뉴클레오티드는 다음을 포함한다:
(a) 서열 번호: 1의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(b)
(i) 서열 번호: 2의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 3의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iii) 서열 번호: 30의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iv) 서열 번호: 31의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(v) 서열 번호: 36의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(vi) 서열 번호: 40의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(vii) 서열 번호: 59의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(viii) 서열 번호: 76의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ix) 서열 번호: 80의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(x) 서열 번호: 81의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(xi) 서열 번호: 92의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 및/또는
(xii) 서열 번호: 93의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(c)
(i) 서열 번호: 40의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 60의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iii) 서열 번호: 68의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iv) 서열 번호: 70의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 및/또는
(v) 서열 번호: 86의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(d)
(i) 서열 번호: 68의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 69의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 및/또는
(iii) 서열 번호: 70의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(e)
(i) 서열 번호: 4의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 5의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iii) 서열 번호: 6의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iv) 서열 번호: 56의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(v) 서열 번호: 57의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(vi) 서열 번호: 58의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(vii) 서열 번호: 91의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(viii) 서열 번호: 94의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 및/또는
(ix) 서열 번호: 95의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(f)
(i) 서열 번호: 20의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 37의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iii) 서열 번호: 38의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iv) 서열 번호: 44의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(v) 서열 번호: 46의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(g)
서열 번호: 12의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(h)
(i) 서열 번호: 7의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 8의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iii) 서열 번호: 96의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iv) 서열 번호: 98의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 및/또는
(v) 서열 번호: 99의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(i)
(i) 서열 번호: 22의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 39의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iii) 서열 번호: 62의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iv) 서열 번호: 74의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(v) 서열 번호: 75의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(vi) 서열 번호: 87의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(vii) 서열 번호: 88의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(viii) 서열 번호: 89의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 및/또는
(ix) 서열 번호: 90의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(i)
(i) 서열 번호: 82의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 83의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iii) 서열 번호: 84의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 및/또는
(iv) 서열 번호: 85의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(k)
(i) 서열 번호: 13의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 14의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iii) 서열 번호: 72의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 및/또는
(iv) 서열 번호: 73의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(l)
(i) 서열 번호: 34의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 35의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iii) 서열 번호: 77의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iv) 서열 번호: 78의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 및/또는
(v) 서열 번호: 79의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(m)
서열 번호: 51의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
및/또는
(n)
(i) 서열 번호: 9의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 10의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iii) 서열 번호: 11의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iv) 서열 번호: 15의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(v) 서열 번호: 16의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(vi) 서열 번호: 17의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(vii) 서열 번호: 18의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(viii) 서열 번호: 27의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ix) 서열 번호: 32의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(x) 서열 번호: 33의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(xi) 서열 번호: 41의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(xii) 서열 번호: 49의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(xiii) 서열 번호: 50의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(xiv) 서열 번호: 52의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(xv) 서열 번호: 53의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(xvi) 서열 번호: 61의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 및/또는
(xvii) 서열 번호: 63의 적어도 5개 (예를 들면, 적어도 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열.
일부 구체예에서, 서열 동일성은 서열 번호: 1-12, 13-18, 20, 22, 27, 30-41, 44, 46, 49-53, 56-63, 68-70, 72-92, 또는 94-99 중에서 하나의 적어도 10개 (예를 들면, 적어도 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 결정된다. 일부 구체예에서, 서열 동일성은 서열 번호: 1-12, 13-18, 20, 22, 27, 30-41, 44, 46, 49-53, 56-63, 68-70, 72-92, 또는 94-99 중에서 하나의 적어도 15개 (예를 들면, 적어도 15, 16, 17, 18, 19, 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 결정된다. 일부 구체예에서, 서열 동일성은 서열 번호: 1-12, 13-18, 20, 22, 27, 30-41, 44, 46, 49-53, 56-63, 68-70, 72-92, 또는 94-99 중에서 하나의 적어도 20개 (예를 들면, 적어도 20, 21, 또는 22개)의 연속 뉴클레오티드에 걸쳐 결정된다. 일부 구체예에서, 서열 동일성은 서열 번호: 1-12, 13-18, 20, 22, 27, 30-41, 44, 46, 49-53, 56-63, 68-70, 72-92, 또는 94-99 중에서 하나의 전장에 걸쳐 결정된다.
일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 68의 핵산 서열을 포함한다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 68 및 649의 핵산 서열을 포함한다. 일부 구체예에서, 폴리뉴클레오티드는 5'에서 3'로, 서열 번호: 68, 758 및 649의 핵산 서열을 포함한다. 일부 구체예에서, 폴리뉴클레오티드는 5'에서 3'로, 서열 번호: 649, 758 및 68의 핵산 서열을 포함한다. 일부 구체예에서, 폴리뉴클레오티드는 5'에서 3'로, 서열 번호: 649, 758 및 68의 핵산 서열을 포함한다. 일부 구체예에서, 폴리뉴클레오티드는 5'에서 3'로, 서열 번호: 752, 68, 758, 649 및 753의 핵산 서열을 포함한다. 일부 구체예에서, 폴리뉴클레오티드는 5'에서 3'로, 서열 번호: 752, 649, 758, 68 및 753의 핵산 서열을 포함한다.
일부 구체예에서, 폴리뉴클레오티드는 Grik2 mRNA의 비코딩 서열에 혼성화한다. 일부 구체예에서, 비코딩 서열은 Grik2 mRNA의 5' 비번역 영역 (UTR)을 포함한다. 일부 구체예에서, 5' UTR은 서열 번호: 126의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 폴리뉴클레오티드에 의해 인코딩된다. 일부 구체예에서, 비코딩 서열은 Grik2 mRNA의 3' UTR을 포함한다. 일부 구체예에서, 3' UTR은 서열 번호: 127의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 폴리뉴클레오티드에 의해 인코딩된다.
일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 115-681의 핵산 서열 중에서 하나에 혼성화한다.
일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는다.
일부 구체예에서, 폴리뉴클레오티드는 안티센스 올리고뉴클레오티드 (ASO)이다. 일부 구체예에서, ASO는 짧은 간섭 RNA (siRNA), 짧은 헤어핀 RNA (shRNA), 마이크로RNA (miRNA), 또는 shRNA 적응 마이크로RNA (shmiRNA)이다.
일부 구체예에서, 폴리뉴클레오티드는 19-21개 뉴클레오티드 사이에 있다. 일부 구체예에서, 폴리뉴클레오티드는 19개 뉴클레오티드이다. 일부 구체예에서, 폴리뉴클레오티드는 20개 뉴클레오티드이다. 일부 구체예에서, 폴리뉴클레오티드는 21개 뉴클레오티드이다.
일부 구체예에서, Grik2 mRNA는 서열 번호: 115, 서열 번호: 116, 서열 번호: 117, 서열 번호: 118, 서열 번호: 119, 서열 번호: 120, 서열 번호: 121, 서열 번호: 122, 서열 번호: 123, 또는 서열 번호: 124의 핵산 서열에 의해 인코딩된다.
일부 구체예에서, 폴리뉴클레오티드는 세포에서 Gluk2 단백질의 수준을 감소시킬 수 있다. 일부 구체예에서, 폴리뉴클레오티드는 세포에서 GluK2 단백질의 수준을 적어도 10%, 적어도 15%, 적어도 20%, 적어도 25%, 적어도 30%, 적어도 35%, 적어도 40%, 적어도 45%, 적어도 50%, 적어도 55%, 적어도 60%, 적어도 65%, 적어도 70%, 또는 적어도 75% 감소시킨다. 일부 구체예에서, 세포는 뉴런이다. 일부 구체예에서, 뉴런은 해마 뉴런이다. 일부 구체예에서, 해마 뉴런은 치상 과립 세포 (DGC)이다.
앞서 말한 양상의 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 1-771의 서열 중에서 하나와 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 68의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 68 및 649의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다.
다른 양상에서, 본원 발명은 앞서 말한 양상과 구체예의 폴리뉴클레오티드를 포함하는 벡터를 제공한다. 일부 구체예에서, 벡터는 복제 결함성이다. 일부 구체예에서, 복제 결함성 벡터는 비리온 합성, 복제 및 포장에 필요한 유전자의 하나 이상의 코딩 영역을 결여하는 벡터이다. 일부 구체예에서, 벡터는 포유류, 세균, 또는 바이러스 벡터이다. 일부 구체예에서, 벡터는 발현 벡터이다.
일부 구체예에서, 바이러스 벡터는 아데노 관련 바이러스 (AAV), 레트로바이러스, 아데노바이러스, 파보바이러스, 코로나바이러스, 음성 가닥 RNA 바이러스, 오르토믹소바이러스, 랍도바이러스, 파라믹소바이러스, 양성 가닥 RNA 바이러스, 피코르나바이러스, 알파바이러스, 이중 가닥 DNA 바이러스, 헤르페스바이러스, 엡스타인 바르 바이러스, 시토메갈로바이러스, 계두바이러스, 그리고 카나리아두바이러스로 구성된 군에서 선택된다. 일부 구체예에서, 벡터는 AAV 벡터이다. 일부 구체예에서, AAV 벡터는 AAV9 또는 AAVrh10 벡터이다.
일부 구체예에서, 벡터는 본원에서 참조로서 편입되는 U.S. 특허가출원 번호: 63/050,742의 표 9 또는 표 10에서 규정된 서열 중에서 하나를 내포하는 발현 카세트를 포함한다.
일부 구체예에서, 앞서 말한 양상의 벡터는 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 벡터는 서열 번호: 1-771의 서열 중에서 하나 이상과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 벡터는 서열 번호: 68의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 벡터는 서열 번호: 68 및 649의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다.
다른 양상에서, 본원 발명은 표 4에서 설명된 표적 서열로 구성된 군에서 선택되는 Grik2 mRNA 표적 서열 또는 서열 번호: 164-681 중에서 하나, 또는 표 4에서 설명된 상응하는 표적 서열 또는 서열 번호: 164-681 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에 완전히 또는 실제적으로 상보적인 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나 또는 거기에 85%까지 또는 그 초과의 서열 동일성을 갖는 이의 변이체)를 포함하는 발현 카세트를 제공한다.
다른 양상에서, 본원 발명은 표 4에서 설명된 표적 서열로 구성된 군에서 선택되는 Grik2 mRNA 표적 서열 또는 서열 번호: 164-681 중에서 하나, 또는 표 4에서 설명된 상응하는 표적 서열 또는 서열 번호: 164-681 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에 완전히 또는 실제적으로 상보적인 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나 또는 거기에 85%까지 또는 그 초과의 서열 동일성을 갖는 이의 변이체)를 포함하는 발현 카세트를 제공한다.
다른 양상에서, 본원 발명은 표 4에서 설명된 표적 서열로 구성된 군에서 선택되는 Grik2 mRNA 표적 서열 또는 서열 번호: 164-681 중에서 하나, 또는 표 4에서 설명된 상응하는 표적 서열 또는 서열 번호: 164-681 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에 완전히 또는 실제적으로 상보적인 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 CAG 프로모터 (예를 들면, 서열 번호: 737 또는 거기에 85%까지 또는 그 초과의 서열 동일성을 갖는 이의 변이체)를 포함하는 발현 카세트를 제공한다.
다른 양상에서, 본원 발명은 표 4에서 설명된 표적 서열로 구성된 군에서 선택되는 Grik2 mRNA 표적 서열 또는 서열 번호: 164-681 중에서 하나, 또는 표 4에서 설명된 상응하는 표적 서열 또는 서열 번호: 164-681 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에 완전히 또는 실제적으로 상보적인 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 CBA 프로모터 (예를 들면, 서열 번호: 738 또는 거기에 85%까지 또는 그 초과의 서열 동일성을 갖는 이의 변이체)를 포함하는 발현 카세트를 제공한다.
다른 양상에서, 본원 발명은 표 4에서 설명된 표적 서열로 구성된 군에서 선택되는 Grik2 mRNA 표적 서열 또는 서열 번호: 164-681 중에서 하나, 또는 표 4에서 설명된 상응하는 표적 서열 또는 서열 번호: 164-681 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에 완전히 또는 실제적으로 상보적인 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 U6 프로모터 (예를 들면, 서열 번호: 728-733 중에서 하나 또는 거기에 85%까지 또는 그 초과의 서열 동일성을 갖는 이의 변이체)를 포함하는 발현 카세트를 제공한다.
다른 양상에서, 본원 발명은 표 4에서 설명된 표적 서열로 구성된 군에서 선택되는 Grik2 mRNA 표적 서열 또는 서열 번호: 164-681 중에서 하나, 또는 표 4에서 설명된 상응하는 표적 서열 또는 서열 번호: 164-681 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에 완전히 또는 실제적으로 상보적인 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 H1 프로모터 (예를 들면, 서열 번호: 734 또는 거기에 85%까지 또는 그 초과의 서열 동일성을 갖는 이의 변이체)를 포함하는 발현 카세트를 제공한다.
다른 양상에서, 본원 발명은 표 4에서 설명된 표적 서열로 구성된 군에서 선택되는 Grik2 mRNA 표적 서열 또는 서열 번호: 164-681 중에서 하나, 또는 표 4에서 설명된 상응하는 표적 서열 또는 서열 번호: 164-681 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에 완전히 또는 실제적으로 상보적인 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 7SK 프로모터 (예를 들면, 서열 번호: 746 또는 거기에 85%까지 또는 그 초과의 서열 동일성을 갖는 이의 변이체)를 포함하는 발현 카세트를 제공한다.
다른 양상에서, 본원 발명은 서열 번호: 1-100 중에서 하나, 또는 서열 번호: 1-100 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체로 구성된 군에서 선택되는 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나 또는 거기에 85%까지 또는 그 초과의 서열 동일성을 갖는 이의 변이체)를 포함하는 발현 카세트를 제공한다.
다른 양상에서, 본원 발명은 서열 번호: 1-100 중에서 하나, 또는 서열 번호: 1-100 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체로 구성된 군에서 선택되는 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나 또는 거기에 85%까지 또는 그 초과의 서열 동일성을 갖는 이의 변이체)를 포함하는 발현 카세트를 제공한다.
다른 양상에서, 본원 발명은 서열 번호: 1-100 중에서 하나, 또는 서열 번호: 1-100 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체로 구성된 군에서 선택되는 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 CAG 프로모터 (예를 들면, 서열 번호: 737 또는 거기에 85%까지 또는 그 초과의 서열 동일성을 갖는 이의 변이체)를 포함하는 발현 카세트를 제공한다.
다른 양상에서, 본원 발명은 서열 번호: 1-100 중에서 하나, 또는 서열 번호: 1-100 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체로 구성된 군에서 선택되는 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 CBA 프로모터 (예를 들면, 서열 번호: 738 또는 거기에 85%까지 또는 그 초과의 서열 동일성을 갖는 이의 변이체)를 포함하는 발현 카세트를 제공한다.
다른 양상에서, 본원 발명은 서열 번호: 1-100 중에서 하나, 또는 서열 번호: 1-100 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체로 구성된 군에서 선택되는 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 U6 프로모터 (예를 들면, 서열 번호: 728-733 중에서 하나 또는 거기에 85%까지 또는 그 초과의 서열 동일성을 갖는 이의 변이체)를 포함하는 발현 카세트를 제공한다.
다른 양상에서, 본원 발명은 서열 번호: 1-100 중에서 하나, 또는 서열 번호: 1-100 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체로 구성된 군에서 선택되는 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 H1 프로모터 (예를 들면, 서열 번호: 734 또는 거기에 85%까지 또는 그 초과의 서열 동일성을 갖는 이의 변이체)를 포함하는 발현 카세트를 제공한다.
다른 양상에서, 본원 발명은 서열 번호: 1-100 중에서 하나, 또는 서열 번호: 1-100 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체로 구성된 군에서 선택되는 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 7SK 프로모터 (예를 들면, 서열 번호: 746 또는 거기에 85%까지 또는 그 초과의 서열 동일성을 갖는 이의 변이체)를 포함하는 발현 카세트를 제공한다. 다른 양상에서, 본원 발명은 표 9에서 설명된 발현 카세트 중 하나에서 선택되는 발현 카세트를 제공한다 (상세한 설명 참조).
다른 양상에서, 본원 발명은 5'에서 3'로, (i) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 가이드 뉴클레오티드 서열을 포함하는 5' 스템 루프 팔; (ii) 루프 영역으로서, 여기서 상기 루프 영역은 마이크로RNA 루프 서열을 포함하고; (iii) 상기 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 승객 뉴클레오티드 서열을 포함하는 3' 스템 루프 팔을 포함하는 스템 루프 서열을 내포하는 뉴클레오티드 서열을 포함하는 발현 카세트를 제공한다.
다른 양상에서, 본원 발명은 5'에서 3'로, (a) (i) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 가이드 뉴클레오티드 서열을 포함하는 5' 스템 루프 팔; (ii) 루프 영역으로서, 여기서 상기 루프 영역은 마이크로RNA 루프 서열을 포함하고; (iii) 상기 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 승객 뉴클레오티드 서열을 포함하는 3' 스템 루프 팔을 포함하는 스템 루프 서열, (b) 상기 가이드 서열의 5'에 위치된 첫 번째 측부 영역; 및 (c) 상기 승객 서열의 3'에 위치된 두 번째 측부 영역을 내포하는 뉴클레오티드 서열을 포함하는 발현 카세트를 제공한다.
일부 구체예에서, 앞서 말한 양상의 발현 카세트는 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 발현 카세트는 서열 번호: 1-771의 서열 중에서 하나 이상과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 발현 카세트는 서열 번호: 68의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 발현 카세트는 서열 번호: 68 및 649의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 발현 카세트는 서열 번호: 68 및 649의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다.
다른 양상에서, 본원 발명은 5'에서 3'로', (i) 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 승객 뉴클레오티드 서열을 포함하는 5' 스템 루프 팔; (ii) 루프 영역으로서, 여기서 상기 루프 영역은 마이크로RNA 루프 서열을 포함하고; (iii) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 가이드 뉴클레오티드 서열을 포함하는 3' 스템 루프 팔을 포함하는 스템 루프 서열을 포함하는 뉴클레오티드 서열을 포함하는 발현 카세트를 제공한다. 일부 구체예에서, 발현 카세트는 5'에서 3'로', (i) 두 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 두 번째 승객 뉴클레오티드 서열을 포함하는 두 번째 5' 스템 루프 팔; (ii) 두 번째 루프 영역으로서, 여기서 상기 두 번째 루프 영역은 두 번째 마이크로RNA 루프 서열을 포함하고; (iii) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 두 번째 가이드 뉴클레오티드 서열을 포함하는 두 번째 3' 스템 루프 팔을 포함하는 두 번째 스템 루프 서열을 더욱 포함한다. 일부 구체예에서, 첫 번째 스템 루프 서열 및 두 번째 스템 루프 서열은 동일하다. 일부 구체예에서, 첫 번째 스템 루프 서열 및 두 번째 스템 루프 서열은 상이하다.
다른 양상에서, 본원 발명은 (a) 5'에서 3'로, (i) 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 승객 뉴클레오티드 서열을 포함하는 5' 스템 루프 팔; (ii) 루프 영역으로서, 여기서 상기 루프 영역은 마이크로RNA 루프 서열을 포함하고; (iii) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 가이드 뉴클레오티드 서열을 포함하는 3' 스템 루프 팔을 포함하는 스템 루프 서열; (b) 상기 가이드 서열의 5'에 위치된 첫 번째 측부 영역; 및 (c) 상기 승객 서열의 3'에 위치된 두 번째 측부 영역을 포함하는 뉴클레오티드 서열을 포함하는 발현 카세트를 제공한다. 일부 구체예에서, 발현 카세트는 (a) 5'에서 3'로', (i) 두 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 두 번째 승객 뉴클레오티드 서열을 포함하는 두 번째 5' 스템 루프 팔; (ii) 두 번째 루프 영역으로서, 여기서 상기 두 번째 루프 영역은 두 번째 마이크로RNA 루프 서열을 포함하고; (iii) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 두 번째 가이드 뉴클레오티드 서열을 포함하는 두 번째 3' 스템 루프 팔을 포함하는 두 번째 스템 루프 서열; (b) 상기 두 번째 가이드 서열의 5'에 위치된 세 번째 측부 영역; 및 (c) 상기 두 번째 승객 서열의 3'에 위치된 네 번째 측부 영역을 더욱 포함한다. 일부 구체예에서, 첫 번째 스템 루프 서열 및 두 번째 스템 루프 서열은 동일하다. 일부 구체예에서, 첫 번째 스템 루프 서열 및 두 번째 스템 루프 서열은 상이하다.
앞서 말한 양상과 구체예의 일부 구체예에서, 첫 번째 측부 영역은 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 폴리뉴클레오티드를 포함한다. 일부 구체예에서, 두 번째 측부 영역은 서열 번호: 753, 755, 757, 760, 763, 766 또는 769 중에서 하나의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 폴리뉴클레오티드를 포함한다.
일부 구체예에서, 첫 번째 측부 영역은 5' 스페이서 서열 및 5' 측부 서열을 포함한다. 일부 구체예에서, 두 번째 측부 영역은 3' 스페이서 서열 및 3' 측부 서열을 포함한다.
일부 구체예에서, 마이크로RNA 루프 서열은 miR-30, miR-155, miR-218-1, 또는 miR-124-3 서열이다. 일부 구체예에서, 마이크로RNA 루프 서열은 서열 번호: 758, 761, 764, 767 또는 770 중에서 하나의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 폴리뉴클레오티드를 포함한다.
일부 구체예에서, 발현 카세트는 U6 프로모터, H1 프로모터, 7SK 프로모터, 아포지질단백질 E-인간 알파 1-항트립신 (ApoE-hAAT) 프로모터, CAG 프로모터, CBA 프로모터, CK8 프로모터, mU1a 프로모터, 신장 인자 1α (EF1α) 프로모터, 단순 헤르페스 바이러스 (HSV) 프로모터 티록신 결합 글로불린 (TBG) 프로모터, 시냅신 프로모터 (SYN), RNA 결합 Fox-1 동족체 3 (RBFOX3) 프로모터, 칼슘/칼모듈린 의존성 단백질 키나아제 II (CaMKII) 프로모터, 뉴런 특이적 에놀라아제 (NSE) 프로모터, 혈소판 유래 성장 인자 아단위 β (PDGFβ) 프로모터, 소포성 글루타민산염 전달체 (VGAT) 프로모터, 소마토스타틴 (SST) 프로모터, 신경펩티드 Y (NPY) 프로모터, 혈관작용 장관 펩티드 (VIP) 프로모터, 파브알부민 (PV) 프로모터, 글루타민산염 탈카르복실화효소 65 (GAD65) 프로모터, 글루타민산염 탈카르복실화효소 67 (GAD67) 프로모터, 도파민 수용체 D1 (DRD1) 프로모터, 도파민 수용체 D2 (DRD2) 프로모터, 보체 C1q 유사 2 (C1QL2) 프로모터, 프로오피오멜라노코르틴 (POMC) 프로모터, 프로스페로 호메오복스 1 (PROX1) 프로모터, 미소관 관련 단백질 1B (MAP1B) 프로모터, 그리고 튜불린 알파 1 (T-α1/TUBA3) 프로모터로 구성된 군에서 선택되는 프로모터를 포함한다. 일부 구체예에서, 발현 카세트는 SYN 프로모터 (예를 들면, 예컨대 인간 SYN 프로모터, 예를 들면, 서열 번호: 682-685 및 790 중에서 하나, 또는 서열 번호: 682-682 및 790 중에서 하나의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)를 포함한다. 일부 구체예에서, 발현 카세트는 CAMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나, 또는 서열 번호: 687-691 및 802 중에서 하나의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)를 포함한다. 일부 구체예에서, 발현 카세트는 C1QL2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791, 또는 서열 번호: 719 또는 서열 번호: 791의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)를 포함한다. 일부 구체예에서, 프로모터는 2개 이상의 스템 루프 서열에 작동가능하게 연결된다. 일부 구체예에서, 프로모터는 2개의 스템 루프 서열 (예를 들면, 벡터 내에 나란히 존재하는 2개의 스템 루프 서열)에 작동가능하게 연결된다.
일부 구체예에서, 발현 카세트는 서열 번호: 775, 777, 779, 781, 783-788, 796, 798-801, 803, 805, 807, 809, 811, 813, 817, 819 및 821 중에서 하나의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 폴리뉴클레오티드를 포함한다. 일부 구체예에서, 발현 카세트는 서열 번호: 804, 806, 808, 810, 812, 814, 818, 820 및 822 중에서 하나의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 벡터 내로 통합된다.
다른 양상에서, 본원 발명은 5'에서 3'로, 다음을 포함하는 발현 카세트를 제공한다: (a) 첫 번째 프로모터 서열 (예를 들면, 본원에서 개시된 프로모터 서열 중에서 하나, 예컨대 예를 들면, 표 5에서 개시된 것들, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나), 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); (b) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 첫 번째 가이드 뉴클레오티드 서열로서, 여기서 상기 첫 번째 가이드 뉴클레오티드 서열은 첫 번째 프로모터에 작동가능하게 연결되고; (c) 임의적으로, 두 번째 프로모터 서열 (예를 들면, 본원에서 개시된 프로모터 서열 중에서 하나, 예컨대 예를 들면, 표 5에서 개시된 것들, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나), 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); (b) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 두 번째 가이드 뉴클레오티드 서열로서, 여기서 임의적으로, 상기 두 번째 가이드 뉴클레오티드 서열은 두 번째 프로모터에 작동가능하게 연결된다. 일부 구체예에서, 첫 번째 가이드 서열 및/또는 두 번째 가이드 서열은 G9 서열 (서열 번호: 68) 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이다. 일부 구체예에서, 첫 번째 가이드 서열 및/또는 두 번째 가이드 서열은 GI 서열 (서열 번호: 77) 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이다. 일부 구체예에서, 첫 번째 가이드 서열 및/또는 두 번째 가이드 서열은 MW 서열 (서열 번호: 80) 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이다. 일부 구체예에서, 첫 번째 가이드 서열 및/또는 두 번째 가이드 서열은 MU 서열 (서열 번호: 96) 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이다. 일부 구체예에서, 첫 번째 가이드 서열은 G9 서열 (서열 번호: 68)이거나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이고, 그리고 두 번째 가이드 서열은 GI 서열 (서열 번호: 77)이거나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이다. 일부 구체예에서, 첫 번째 가이드 서열은 G9 서열 (서열 번호: 68)이거나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이고, 그리고 두 번째 가이드 서열은 MW 서열 (서열 번호: 80)이거나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이다. 일부 구체예에서, 첫 번째 가이드 서열은 GI 서열 (서열 번호: 77)이거나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이고, 그리고 두 번째 가이드 서열은 G9 서열 (서열 번호: 68)이거나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이다. 일부 구체예에서, 첫 번째 가이드 서열은 GI 서열 (서열 번호: 77)이거나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이고, 그리고 두 번째 가이드 서열은 MW 서열 (서열 번호: 80)이거나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이다.
일부 구체예에서, 발현 카세트는 서열 번호: 785-788 중에서 하나의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 폴리뉴클레오티드를 포함한다.
일부 구체예에서, 첫 번째 프로모터는 SYN 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나 또는 거기에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이고, 그리고 임의적으로, 두 번째 프로모터는 CAMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나 또는 거기에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이다.
앞서 말한 양상의 일부 구체예에서, 발현 카세트는 첫 번째 가이드 뉴클레오티드 서열에 상보적이거나 또는 실질적으로 상보적인 첫 번째 승객 뉴클레오티드 서열을 더욱 포함하고, 여기서 첫 번째 승객 뉴클레오티드 서열은 첫 번째 가이드 뉴클레오티드 서열에 비하여 5' 또는 3'에 위치된다.
앞서 말한 양상의 일부 구체예에서, 발현 카세트는 두 번째 가이드 뉴클레오티드 서열에 상보적이거나 또는 실질적으로 상보적인 두 번째 승객 뉴클레오티드 서열을 더욱 포함하고, 여기서 두 번째 승객 뉴클레오티드 서열은 두 번째 가이드 뉴클레오티드 서열에 비하여 5' 또는 3'에 위치된다.
앞서 말한 양상의 일부 구체예에서, 발현 카세트는 첫 번째 가이드 서열에 비하여 5' 에 위치된 첫 번째 5' 측부 영역 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 및 768 중에서 하나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)을 더욱 포함한다.
앞서 말한 양상의 일부 구체예에서, 발현 카세트는 첫 번째 가이드 서열에 비하여 3' 에 위치된 첫 번째 3' 측부 영역을 더욱 포함한다.
앞서 말한 양상의 일부 구체예에서, 발현 카세트는 두 번째 가이드 서열에 비하여 5' 에 위치된 두 번째 5' 측부 영역을 더욱 포함한다.
앞서 말한 양상의 일부 구체예에서, 발현 카세트는 두 번째 가이드 서열에 비하여 3' 에 위치된 두 번째 3' 측부 영역을 더욱 포함한다.
앞서 말한 양상의 일부 구체예에서, 발현 카세트는 첫 번째 가이드 서열 및 첫 번째 승객 서열 사이에 위치된 첫 번째 루프 영역을 더욱 포함하고, 여기서 첫 번째 루프 영역은 첫 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 및 770 중에서 하나 또는 거기에 1개, 2개 또는 3개의 뉴클레오티드 변화를 갖는 이의 변이체)을 포함한다.
앞서 말한 양상의 일부 구체예에서, 발현 카세트는 두 번째 가이드 서열 및 두 번째 승객 서열 사이에 위치된 두 번째 루프 영역을 더욱 포함하고, 여기서 두 번째 루프 영역은 두 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 및 770 중에서 하나 또는 거기에 1개, 2개 또는 3개의 뉴클레오티드 변화를 갖는 이의 변이체)을 포함한다.
다른 양상에서, 본원 발명은 5'에서 3'로, 다음을 포함하는 뉴클레오티드 서열을 포함하는 발현 카세트를 제공한다:
(a) 첫 번째 프로모터 서열 (예를 들면, 본원에서 개시된 프로모터 서열 중에서 하나, 예컨대 예를 들면, 표 5에서 개시된 것들, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나), 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체);
(b) 첫 번째 승객 뉴클레오티드 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 및 768 중에서 하나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)의 5'에 위치된 첫 번째 5' 측부 영역;
(c) 첫 번째 스템 루프 서열로서, 5'에서 3'로:
(i) 첫 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 첫 번째 승객 뉴클레오티드 서열을 포함하는 첫 번째 5′ 스템 루프 팔;
(ii) 첫 번째 루프 영역으로서, 여기서 상기 첫 번째 루프 영역은 첫 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 및 770 중에서 하나 또는 거기에 1개, 2개 또는 3개의 뉴클레오티드 변화를 갖는 이의 변이체)을 포함하고;
(iii) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 첫 번째 가이드 뉴클레오티드 서열을 포함하는 첫 번째 3′ 스템 루프 팔을 포함하는 첫 번째 스템 루프 서열;
(d) 상기 첫 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 및 769 중에서 하나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)의 3'에 위치된 첫 번째 3' 측부 영역;
(e) 임의적으로, 두 번째 프로모터 서열 (예를 들면, 본원에서 개시된 프로모터 서열 중에서 하나, 예컨대 예를 들면, 표 5에서 개시된 것들, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나), 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체);
(f) 두 번째 승객 뉴클레오티드 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 및 768 중에서 하나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)의 5'에 위치된 두 번째 5' 측부 영역;
(g) 두 번째 스템 루프 서열로서, 5'에서 3'로:
(i) 두 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 두 번째 승객 뉴클레오티드 서열을 포함하는 두 번째 5′ 스템 루프 팔;
(ii) 두 번째 루프 영역으로서, 여기서 상기 두 번째 루프 영역은 두 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 및 770 중에서 하나 또는 거기에 1개, 2개 또는 3개의 뉴클레오티드 변화를 갖는 이의 변이체)을 포함하고;
(iii) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 두 번째 가이드 뉴클레오티드 서열을 포함하는 두 번째 3′ 스템 루프 팔을 포함하는 두 번째 스템 루프 서열;
그리고
(h) 상기 두 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 및 769 중에서 하나)의 3'에 위치된 두 번째 3' 측부 영역.
일부 구체예에서, 발현 카세트는 서열 번호: 785, 787 및 788 중에서 하나의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 폴리뉴클레오티드를 포함한다.
다른 양상에서, 본원 발명은 5'에서 3'로, 다음을 포함하는 뉴클레오티드 서열을 포함하는 발현 카세트를 제공한다:
(a) 첫 번째 프로모터 서열 (예를 들면, 본원에서 개시된 프로모터 서열 중에서 하나, 예컨대 예를 들면, 표 5에서 개시된 것들, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나), 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체);
(b) 첫 번째 승객 뉴클레오티드 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 및 768 중에서 하나)의 5'에 위치된 첫 번째 5' 측부 영역;
(c) 첫 번째 스템 루프 서열로서, 5'에서 3'로:
(i) 첫 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 첫 번째 승객 뉴클레오티드 서열을 포함하는 첫 번째 5′ 스템 루프 팔;
(ii) 첫 번째 루프 영역으로서, 여기서 상기 첫 번째 루프 영역은 첫 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 및 770 중에서 하나)을 포함하고;
(iii) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 첫 번째 가이드 뉴클레오티드 서열을 포함하는 첫 번째 3′ 스템 루프 팔을 포함하는 첫 번째 스템 루프 서열;
(d) 상기 첫 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 및 769 중에서 하나)의 3'에 위치된 첫 번째 3' 측부 영역;
(e) 임의적으로, 두 번째 프로모터 서열 (예를 들면, 본원에서 개시된 프로모터 서열 중에서 하나, 예컨대 예를 들면, 표 5에서 개시된 것들, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나), 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체);
(f) 두 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 및 768 중에서 하나)의 5'에 위치된 두 번째 5' 측부 영역;
(g) 두 번째 스템 루프 서열로서, 5'에서 3'로:
(i) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 가이드 뉴클레오티드 서열을 포함하는 두 번째 5′ 스템 루프 팔;
(ii) 두 번째 루프 영역으로서, 여기서 상기 두 번째 루프 영역은 두 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 및 770 중에서 하나)을 포함하고;
(iii) 두 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 두 번째 승객 뉴클레오티드 서열을 포함하는 두 번째 3′ 스템 루프 팔을 포함하는 두 번째 스템 루프 서열;
그리고
(h) 상기 두 번째 승객 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 및 769 중에서 하나)의 3'에 위치된 두 번째 3' 측부 영역.
다른 양상에서, 본원 발명은 5'에서 3'로, 다음을 포함하는 뉴클레오티드 서열을 포함하는 발현 카세트를 제공한다:
(a) 첫 번째 프로모터 서열 (예를 들면, 본원에서 개시된 프로모터 서열 중에서 하나, 예컨대 예를 들면, 표 5에서 개시된 것들, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나), 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체);
(b) 첫 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 및 768 중에서 하나)의 5'에 위치된 첫 번째 5' 측부 영역;
(c) 첫 번째 스템 루프 서열로서, 5'에서 3'로:
(i) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 첫 번째 가이드 뉴클레오티드 서열을 포함하는 첫 번째 5′ 스템 루프 팔;
(ii) 첫 번째 루프 영역으로서, 여기서 상기 첫 번째 루프 영역은 첫 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 및 770 중에서 하나)을 포함하고;
(iii) 첫 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 첫 번째 승객 뉴클레오티드 서열을 포함하는 첫 번째 3′ 스템 루프 팔을 포함하는 첫 번째 스템 루프 서열;
(d) 상기 첫 번째 승객 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 및 769 중에서 하나)의 3'에 위치된 첫 번째 3' 측부 영역;
(e) 임의적으로, 두 번째 프로모터 서열 (예를 들면, 본원에서, 예를 들면 표 5에서 개시된 프로모터 서열 중에서 하나, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나), 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체);
(f) 두 번째 승객 뉴클레오티드 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 및 768 중에서 하나)의 5'에 위치된 두 번째 5' 측부 영역;
(g) 두 번째 스템 루프 서열로서, 5'에서 3'로:
(i) 두 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 두 번째 승객 뉴클레오티드 서열을 포함하는 두 번째 5′ 스템 루프 팔;
(ii) 두 번째 루프 영역으로서, 여기서 상기 두 번째 루프 영역은 두 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 및 770 중에서 하나)을 포함하고;
(iii) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 두 번째 가이드 뉴클레오티드 서열을 포함하는 두 번째 3′ 스템 루프 팔을 포함하는 두 번째 스템 루프 서열;
그리고
(h) 상기 두 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 및 769 중에서 하나)의 3'에 위치된 두 번째 3' 측부 영역.
일부 구체예에서, 발현 카세트는 서열 번호: 786의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 폴리뉴클레오티드를 포함한다.
다른 양상에서, 본원 발명은 5'에서 3'로, 다음을 포함하는 뉴클레오티드 서열을 포함하는 발현 카세트를 제공한다:
(a) 첫 번째 프로모터 서열 (예를 들면, 본원에서 개시된 프로모터 서열 중에서 하나, 예컨대 예를 들면, 표 5에서 개시된 것들, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나), 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체);
(b) 첫 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 및 768 중에서 하나)의 5'에 위치된 첫 번째 5' 측부 영역;
(c) 첫 번째 스템 루프 서열로서, 5'에서 3'로:
(i) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 첫 번째 가이드 뉴클레오티드 서열을 포함하는 첫 번째 5′ 스템 루프 팔;
(ii) 첫 번째 루프 영역으로서, 여기서 상기 첫 번째 루프 영역은 첫 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 및 770 중에서 하나)을 포함하고;
(iii) 첫 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 첫 번째 승객 뉴클레오티드 서열을 포함하는 첫 번째 3′ 스템 루프 팔을 포함하는 첫 번째 스템 루프 서열;
(d) 상기 첫 번째 승객 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 및 769 중에서 하나)의 3'에 위치된 첫 번째 3' 측부 영역;
(e) 임의적으로, 두 번째 프로모터 서열 (예를 들면, 본원에서 개시된 프로모터 서열 중에서 하나, 예컨대 예를 들면, 표 5에서 개시된 것들, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나), 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체);
(f) 두 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 및 768 중에서 하나)의 5'에 위치된 두 번째 5' 측부 영역;
(g) 두 번째 스템 루프 서열로서, 5'에서 3'로:
(i) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 가이드 뉴클레오티드 서열을 포함하는 두 번째 5′ 스템 루프 팔;
(ii) 두 번째 루프 영역으로서, 여기서 상기 두 번째 루프 영역은 두 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 및 770 중에서 하나)을 포함하고;
(iii) 두 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 두 번째 승객 뉴클레오티드 서열을 포함하는 두 번째 3′ 스템 루프 팔을 포함하는 두 번째 스템 루프 서열;
그리고
(h) 상기 두 번째 승객 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 및 769 중에서 하나)의 3'에 위치된 두 번째 3' 측부 영역.
일부 구체예에서, 첫 번째 프로모터 및/또는 임의적으로, 두 번째 프로모터는 U6 프로모터, H1 프로모터, 7SK 프로모터, 아포지질단백질 E-인간 알파 1-항트립신 프로모터, CAG 프로모터, CBA 프로모터, CK8 프로모터, mU1a 프로모터, 신장 인자 1α 프로모터, HSV 프로모터, 티록신 결합 글로불린 프로모터, 시냅신 프로모터, RNA 결합 Fox-1 동족체 3 프로모터, 칼슘/칼모듈린 의존성 단백질 키나아제 II 프로모터, 뉴런 특이적 에놀라아제 프로모터, 혈소판 유래 성장 인자 아단위 β, 소포성 글루타민산염 전달체 프로모터, 소마토스타틴 프로모터, 신경펩티드 Y 프로모터, 혈관작용 장관 펩티드 프로모터, 파브알부민 프로모터, 글루타민산염 탈카르복실화효소 65 프로모터, 글루타민산염 탈카르복실화효소 67 프로모터, 도파민 수용체 D1 프로모터, 도파민 수용체 D2 프로모터, 보체 C1q 유사 2 프로모터, 프로오피오멜라노코르틴 프로모터, 프로스페로 호메오복스 1 프로모터, 미소관 관련 단백질 1B 프로모터, 그리고 튜불린 알파 1 프로모터로 구성된 군에서 선택된다.
일부 구체예에서, 첫 번째 프로모터는 SYN 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나 또는 거기에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이고, 그리고 임의적으로, 두 번째 프로모터는 CAMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나 또는 거기에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이다.
일부 구체예에서, 첫 번째 5' 측부 영역 및/또는 두 번째 5' 측부 영역은 서열 번호: 752, 754, 756, 759, 762, 765 및 768 중에서 하나의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 폴리뉴클레오티드를 포함한다. 일부 구체예에서, 첫 번째 5' 측부 영역 및/또는 두 번째 5' 측부 영역은 752, 754, 756, 759, 762, 765 및 768의 핵산 서열을 갖는 폴리뉴클레오티드를 포함한다.
일부 구체예에서, 첫 번째 3' 측부 영역 및/또는 두 번째 3' 측부 영역은 서열 번호: 753, 755, 757, 760, 763, 766 및 769 중에서 하나의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 폴리뉴클레오티드를 포함한다. 일부 구체예에서, 첫 번째 3' 측부 영역 및/또는 두 번째 3' 측부 영역은 서열 번호: 753, 755, 757, 760, 763, 766 및 769 중에서 하나의 핵산 서열을 갖는 폴리뉴클레오티드를 포함한다.
일부 구체예에서, 첫 번째 마이크로RNA 루프 서열 및/또는 두 번째 마이크로RNA 루프 서열은 miR-30, miR-155, miR-218-1, 또는 miR-124-3 서열이다. 일부 구체예에서, 첫 번째 마이크로RNA 루프 서열 및/또는 두 번째 마이크로RNA 루프 서열은 서열 번호: 758, 761, 764, 767 및 770 중에서 하나의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 폴리뉴클레오티드를 포함한다. 일부 구체예에서, 첫 번째 마이크로RNA 루프 서열 및/또는 두 번째 마이크로RNA 루프 서열은 서열 번호: 758, 761, 764, 767 및 770 중에서 하나의 핵산 서열을 갖는 폴리뉴클레오티드를 포함한다.
일부 구체예에서, 발현 카세트는 상기 발현 카세트의 5' 단부 상에서 5'-반전 말단 반복 (ITR) 서열 및 상기 발현 카세트의 3' 단부 상에서 3'-ITR 서열을 포함한다. 일부 구체예에서, 5'-ITR 및 3' ITR 서열은 AAV2 5'-ITR 및 3' ITR 서열이다. 일부 구체예에서, 5'-ITR 서열은 서열 번호: 746 또는 서열 번호: 747의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 폴리뉴클레오티드를 포함한다. 일부 구체예에서, 5'-ITR 서열은 서열 번호: 746 또는 서열 번호: 747의 핵산 서열을 갖는 폴리뉴클레오티드를 포함한다. 일부 구체예에서, 3'-ITR 서열은 서열 번호: 748, 서열 번호: 749, 또는 서열 번호: 789의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 폴리뉴클레오티드를 포함한다. 일부 구체예에서, 3'-ITR 서열은 서열 번호: 748, 서열 번호: 749, 또는 서열 번호: 789의 핵산 서열을 갖는 폴리뉴클레오티드를 포함한다.
일부 구체예에서, 발현 카세트는 인핸서 서열을 더욱 포함한다. 일부 구체예에서, 인핸서 서열은 서열 번호: 745의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 폴리뉴클레오티드를 포함한다. 일부 구체예에서, 인핸서 서열은 서열 번호: 745의 핵산 서열을 갖는 폴리뉴클레오티드를 포함한다.
일부 구체예에서, 발현 카세트는 인트론 서열을 더욱 포함한다. 일부 구체예에서, 인트론 서열은 서열 번호: 743 또는 서열 번호: 744의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 폴리뉴클레오티드를 포함한다. 일부 구체예에서, 인트론 서열은 서열 번호: 743 또는 서열 번호: 744의 핵산 서열을 갖는 폴리뉴클레오티드를 포함한다.
일부 구체예에서, 발현 카세트는 하나 이상의 폴리아데닐화 신호를 더욱 포함한다. 일부 구체예에서, 하나 이상의 폴리아데닐화 신호는 토끼 베타-글로빈 (RBG) 폴리아데닐화 신호이다. 일부 구체예에서, RBG 폴리아데닐화 신호는 서열 번호: 750, 서열 번호: 751, 또는 서열 번호: 792의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 폴리뉴클레오티드를 포함한다. 일부 구체예에서, RBG 폴리아데닐화 신호는 서열 번호: 750, 서열 번호: 751, 또는 서열 번호: 792의 핵산 서열을 갖는 폴리뉴클레오티드를 포함한다. 일부 구체예에서, 폴리아데닐화 신호는 소 성장 호르몬 (BGH) 폴리아데닐화 신호이다. 일부 구체예에서, BGH 폴리아데닐화 신호는 서열 번호: 793의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 폴리뉴클레오티드를 포함한다. 일부 구체예에서, BGH 폴리아데닐화 신호는 서열 번호: 793의 핵산 서열을 갖는 폴리뉴클레오티드를 포함한다.
일부 구체예에서, 앞서 말한 양상과 구체예의 발현 카세트는 앞서 말한 양상과 구체예의 벡터 내로 통합된다.
일부 구체예에서, 앞서 말한 양상의 발현 카세트는 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 발현 카세트는 서열 번호: 1-771의 서열 중에서 하나 이상과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 발현 카세트는 서열 번호: 68의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다. 일부 구체예에서, 발현 카세트는 서열 번호: 68 및 649의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 포함하지 않는다.
일부 구체예에서, 발현 카세트는 하나 이상 (예를 들면, 1개, 2개, 또는 그 초과)의 스터퍼 서열을 더욱 포함한다. 일부 구체예에서, 하나 이상의 스터퍼 서열은 발현 카세트의 3' 단부에 배치된다. 일부 구체예에서, 하나 이상의 스터퍼 서열은 서열 번호: 815의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는다. 일부 구체예에서, 하나 이상의 스터퍼 서열은 서열 번호: 815의 핵산 서열에 적어도 90% (예를 들면, 적어도 91%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는다. 일부 구체예에서, 하나 이상의 스터퍼 서열은 서열 번호: 815의 핵산 서열에 적어도 95% (예를 들면, 적어도 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는다. 일부 구체예에서, 하나 이상의 스터퍼 서열은 서열 번호: 815의 핵산 서열에 적어도 99% 서열 동일성을 갖는다. 일부 구체예에서, 하나 이상의 스터퍼 서열은 서열 번호: 815의 핵산 서열을 갖는다. 일부 구체예에서, 하나 이상의 스터퍼 서열은 서열 번호: 816의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는다. 일부 구체예에서, 하나 이상의 스터퍼 서열은 서열 번호: 816의 핵산 서열에 적어도 90% (예를 들면, 적어도 91%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는다. 일부 구체예에서, 하나 이상의 스터퍼 서열은 서열 번호: 816의 핵산 서열에 적어도 95% (예를 들면, 적어도 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는다. 일부 구체예에서, 하나 이상의 스터퍼 서열은 서열 번호: 816의 핵산 서열에 적어도 99% 서열 동일성을 갖는다. 일부 구체예에서, 하나 이상의 스터퍼 서열은 서열 번호: 816의 핵산 서열을 갖는다.
다른 양상에서, 본원 발명은 세포에서 Grik2 발현을 저해하는 방법을 제공하는데, 상기 방법은 세포를 앞서 말한 양상과 구체예의 적어도 하나의 폴리뉴클레오티드, 앞서 말한 양상과 구체예의 벡터, 또는 앞서 말한 양상과 구체예의 발현 카세트와 접촉시키는 단계를 포함한다.
일부 구체예에서, 폴리뉴클레오티드는 Grik2 mRNA에 특이적으로 혼성화하고, 그리고 세포에서 Grik2 의 발현을 저해하거나 또는 감소시킨다. 일부 구체예에서, 상기 방법은 세포에서 Grik2 mRNA의 수준을 감소시킨다. 일부 구체예에서, 상기 방법은 Grik2 mRNA에 혼성화할 수 없는 대조 폴리뉴클레오티드로 처리된 세포에서 GluK2 단백질의 수준에 비하여 또는 본원 발명의 폴리뉴클레오티드로 처리되지 않은 세포에 비하여, 세포에서 Grik2 mRNA의 수준을 적어도 10%, 적어도 적어도 15%, 적어도 20%, 적어도 25%, 적어도 30%, 적어도 35%, 적어도 40%, 적어도 45%, 적어도 50%, 적어도 55%, 적어도 60%, 적어도 65%, 적어도 70%, 또는 적어도 75% 감소시킨다. 일부 구체예에서, 상기 방법은 세포에서 GluK2 단백질의 수준을 감소시킨다. 일부 구체예에서, 상기 방법은 Grik2 mRNA에 혼성화할 수 없는 대조 폴리뉴클레오티드로 처리된 세포에서 GluK2 단백질의 수준에 비하여 또는 본원 발명의 폴리뉴클레오티드로 처리되지 않은 세포에 비하여, 세포에서 GluK2 단백질의 수준을 적어도 10%, 적어도 적어도 15%, 적어도 20%, 적어도 25%, 적어도 30%, 적어도 35%, 적어도 40%, 적어도 45%, 적어도 50%, 적어도 55%, 적어도 60%, 적어도 65%, 적어도 70%, 또는 적어도 75% 감소시킨다.
일부 구체예에서, 세포는 뉴런이다. 일부 구체예에서, 뉴런은 해마 뉴런이다. 일부 구체예에서, 해마 뉴런은 DGC이다. 일부 구체예에서, DGC는 일탈적 재발성 이끼 섬유 축삭돌기를 포함한다. 세포는 또한, 유도 만능 줄기 세포 (iPSC)로부터 유래된 뉴런 세포, 예컨대 Grik2를 발현하는 iPSC 유래된 글루타민산성 뉴런일 수 있다.
일부 구체예에서, 앞서 말한 양상의 방법은 서열 번호: 772-774 중에서 하나의 서열의 이용을 포함하지 않는다. 일부 구체예에서, 상기 방법은 서열 번호: 1-771의 서열 중에서 하나 이상과 조합으로 서열 번호: 772-774 중에서 하나의 서열의 이용을 포함하지 않는다. 일부 구체예에서, 상기 방법은 서열 번호: 68의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열의 이용을 포함하지 않는다. 일부 구체예에서, 상기 방법은 서열 번호: 68 및 서열 번호: 649의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열의 이용을 포함하지 않는다.
다른 양상에서, 본원 발명은 치료가 필요한 개체에서 장애를 치료하거나 또는 개선하는 방법을 제공하는데, 상기 방법은 앞서 말한 양상과 구체예의 적어도 하나의 폴리뉴클레오티드, 앞서 말한 양상과 구체예의 벡터, 또는 앞서 말한 양상과 구체예의 발현 카세트 (예를 들면, 서열 번호: 775, 777, 779, 781, 783-788, 796, 798-801, 803, 805, 807, 809, 811, 813, 817, 819 및 821 중에서 하나의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 폴리뉴클레오티드를 포함하는 발현 카세트)를 상기 개체에게 투여하는 단계를 포함한다.
일부 구체예에서, 장애는 뇌전증이다. 일부 구체예에서, 뇌전증은 측두엽 뇌전증 (TLE), 만성 뇌전증 및/또는 난치성 뇌전증이다. 일부 구체예에서, 뇌전증은 TLE이다. 일부 구체예에서, TLE는 편측 TLE (lTLE)이다. 일부 구체예에서, TLE는 내측 TLE (mTLE)이다.
전술된 구체예 중에서 하나 이상, 또는 전술된 구체예 각각에서, 개체는 인간이다.
일부 구체예에서, 앞서 말한 양상의 방법은 서열 번호: 772-774 중에서 하나의 서열의 투여를 포함하지 않는다. 일부 구체예에서, 상기 방법은 서열 번호: 1-771의 서열 중에서 하나 이상과 조합으로 서열 번호: 772-774 중에서 하나의 서열의 투여를 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 68의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열의 투여를 포함하지 않는다. 일부 구체예에서, 폴리뉴클레오티드는 서열 번호: 68 및 서열 번호: 649의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열의 투여를 포함하지 않는다.
다른 양상에서, 본원 발명은 앞서 말한 양상과 구체예의 폴리뉴클레오티드, 앞서 말한 양상과 구체예의 벡터, 또는 앞서 말한 양상과 구체예의 발현 카세트, 그리고 약학적으로 허용되는 운반체, 희석제 또는 부형제를 포함하는 약학적 조성물을 제공한다.
일부 구체예에서, 앞서 말한 양상의 약학적 조성물은 서열 번호: 772-774 중에서 하나의 서열을 갖는 폴리뉴클레오티드를 포함하지 않는다. 일부 구체예에서, 약학적 조성물은 서열 번호: 1-771의 서열 중에서 하나 이상과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 갖는 폴리뉴클레오티드를 포함하지 않는다. 일부 구체예에서, 약학적 조성물은 서열 번호: 68의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 갖는 폴리뉴클레오티드를 포함하지 않는다. 일부 구체예에서, 약학적 조성물은 서열 번호: 68 및 서열 번호: 649의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 갖는 폴리뉴클레오티드를 포함하지 않는다.
다른 양상에서, 본원 발명은 앞서 말한 양상의 약학적 조성물 및 포장 삽입물을 포함하는 키트를 제공한다. 일부 구체예에서, 포장 삽입물은 앞서 말한 양상과 구체예의 방법에서 약학적 조성물의 사용법을 포함한다.
일부 구체예에서, 앞서 말한 양상의 키트는 서열 번호: 772-774 중에서 하나의 서열을 갖는 폴리뉴클레오티드를 포함하지 않는다. 일부 구체예에서, 키트는 서열 번호: 1-771의 서열 중에서 하나 이상과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 갖는 폴리뉴클레오티드를 포함하지 않는다. 일부 구체예에서, 키트는 서열 번호: 68의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 갖는 폴리뉴클레오티드를 포함하지 않는다. 일부 구체예에서, 키트는 서열 번호: 68 및 서열 번호: 649의 서열과 조합으로 서열 번호: 772-774 중에서 하나의 서열을 갖는 폴리뉴클레오티드를 포함하지 않는다.
정의
편의를 위해, 본 명세서, 실시예 및 첨부된 청구항에서 이용된 일부 용어와 관용구의 의미가 아래에 제공된다. 별도로 명시되거나, 또는 맥락으로부터 암시되지 않으면, 하기 용어와 관용구는 아래에 제공된 의미를 포함한다. 이들 정의는 특정한 구체예를 설명하는 데 보조하기 위해 제공되고 청구된 기술을 한정하는 것으로 의도되지 않는다. 별도로 규정되지 않으면, 본원에서 이용된 모든 기술 용어와 과학 용어는 본 기술이 속하는 분야의 당업자에 의해 통상적으로 이해되는 바와 동일한 의미를 갖는다. 만약 당해 분야에서 용어의 용법 및 본원에서 제공된 이의 정의 사이에 명백한 불일치가 있으면, 본 명세서 내에 제공된 정의가 우선할 것이다.
본 출원에서, 만약 맥락으로부터 명백하지 않으면, (i) 용어 "a"는 "적어도 하나"를 의미하는 것으로 이해될 수 있고; (ii) 용어 "또는"은 "및/또는"을 의미하는 것으로 이해될 수 있고; 그리고 (iii) 용어 "포함하는" 및 "포함하는"은 그들만으로 또는 한 가지 이상의 추가 구성요소 또는 단계와 함께 제시되는지에 상관없이, 항목별로 구분된 구성요소 또는 단계를 포괄하는 것으로 이해될 수 있다.
용어 "약"은 언급된 값의 ± 10%인 양을 지칭하고, 그리고 언급된 값의 ± 5% 또는 언급된 값의 ± 2%일 수 있다.
용어 "3' 비번역 영역" 및 "3' UTR"은 mRNA 분자 (예를 들면, Grik2 mRNA)의 종결 코돈에 대하여 3'인 영역을 지칭한다. 3' UTR은 단백질로 번역되지 않지만, mRNA 전사체의 폴리아데닐화, 국지화, 안정화 및/또는 번역 효율에 중요한 조절 서열을 포함한다. 3' UTR에서 조절 서열은 인핸서, 사일런서, AU-풍부한 요소, 폴리-A 꼬리, 종결자, 그리고 마이크로RNA 인식 서열을 포함할 수 있다. 용어 "3' 비번역 영역" 및 "3' UTR"은 또한, mRNA 분자를 인코딩하는 유전자의 상응하는 영역을 지칭할 수 있다.
용어 "5' 비번역 영역" 및 "5' UTR"은 개시 코돈에 대하여 5'인 mRNA 분자 (예를 들면, Grik2 mRNA)의 영역을 지칭한다. 이러한 영역은 번역 개시의 조절에 중요하다. 5' UTR은 전적으로 번역되지 않을 수 있거나 또는 이의 영역 중에서 일부가 일부 생물체에서 번역될 수도 있다. 전사 시작 부위는 5' UTR의 시작을 표시하고 개시 코돈으로부터 1개 뉴클레오티드 앞에서 끝난다. 진핵생물에서, 5' UTR은 개시 코돈을 품는 코자크 공통 서열을 포함한다. 5' UTR은 번역의 조절에 중요한, 상류 개방 해독틀로서 또한 알려져 있는 시스-작용 조절 요소를 포함할 수 있다. 이러한 영역은 또한, 상류 AUG 코돈 및 종결 코돈을 품을 수 있다. 많은 GC 함량을 고려할 때, 5' UTR은 번역의 조절에서 일정한 역할을 수행하는 이차 구조, 예컨대 헤어핀 루프를 형성할 수 있다. 용어 "투여"는 임의의 효과적인 루트에 의해, 치료제 (예를 들면, 본원에서 개시된 바와 같이, Grik2 mRNA에 결합하고 이것의 발현을 저해하는 안티센스 올리고뉴클레오티드 (ASO), 또는 이것을 인코딩하는 벡터)를 개체에게 공급하거나 또는 제공하는 것을 지칭한다. 본원에서 예시적인 투여 루트는 아래에 설명된다 (예를 들면 뇌실내 주사, 척수강내 주사, 뇌실질내 주사, 정맥내 주사, 그리고 정위 주사).
용어 "아데노 관련 바이러스 벡터" 또는 "AAV 벡터"는 AAV1, AAV2, AAV3, AAV4, AAV5, AAV6, AAV7, AAV8, AAV9, AAV10, AAV11, AAV12, AAV13, AAV14, AAV15, AAV16, AAV.rh8, AAV.rh10, AAV.rh20, AAV.rh39, AAV.Rh74, AAV.RHM4-1, AAV.hu37, AAV.Anc80, AAV.Anc80L65, AAV.7m8, AAV.PHP.B, AAV.PHP.EB, AAV2.5, AAV2tYF, AAV3B, AAV.LK03, AAV.HSC1, AAV.HSC2, AAV.HSC3, AAV.HSC4, AAV.HSC5, AAV.HSC6, AAV.HSC7, AAV.HSC8, AAV.HSC9, AAV.HSC10, AAV.HSC11, AAV.HSC12, AAV.HSC13, AAV.HSC14, AAV.HSC15, AAV-TT, AAV-DJ8, 또는 AAV.HSC16을 제한 없이 포함하는, 아데노 관련 바이러스 혈청형으로부터 유래된 벡터를 지칭한다. AAV 벡터는 AAV 야생형 유전자 중에서 하나 이상, 예를 들면, rep 및/또는 cap 유전자가 전체적으로 또는 부분적으로 결실되지만, 기능적 측접 ITR 서열을 유지할 수 있다. 기능적 ITR 서열은 AAV 비리온의 구조, 복제 및 포장을 증진한다. 따라서, AAV 벡터는 바이러스의 복제와 포장을 위해 필요한 적어도 이들 서열 (예를 들면, 기능적 ITR)을 시스로 포함하는 것으로 본원에서 규정된다. ITR은 야생형 폴리뉴클레오티드 서열일 필요가 없고, 그리고 이들 서열이 기능적 구조, 복제 및 포장을 제공하기만 하면, 예를 들면, 뉴클레오티드의 삽입, 결실 또는 치환에 의해 변형될 수 있다. AAV 발현 벡터는 전사의 방향으로 작동가능하게 연결된 구성요소로서, 전사 개시 영역을 포함하는 제어 요소, 관심되는 DNA (예를 들면, 본원 발명의 ASO 작용제를 인코딩하는 폴리뉴클레오티드) 및 전사 종결 영역을 적어도 제공하는 공지된 기술을 이용하여 작제된다.
용어 "아데노 관련 바이러스 반전 말단 반복" 및 "AAV ITR"은 DNA 복제의 기점으로서 및 바이러스에 대한 포장 신호로서 시스로 함께 기능하는, AAV 유전체의 각 단부에 측접하는 당해 분야에서 인정된 영역을 지칭한다. AAV ITR은 AAV rep 코딩 영역과 함께, 2개의 측접하는 ITR 사이에 삽입된 폴리뉴클레오티드 서열의 효율적인 절제 및 포유류 유전체 내로의 통합을 제공한다. AAV ITR 영역의 폴리뉴클레오티드 서열은 알려져 있다. 본원에서 이용된 바와 같이, "AAV ITR"은 야생형 폴리뉴클레오티드 서열을 반드시 포함하는 것은 아니며, 예를 들면, 뉴클레오티드의 삽입, 결실 또는 치환에 의해 변형될 수 있다. 추가적으로, AAV ITR은 그 중에서도 특히, AAV1, AAV2, AAV3, AAV4, AAV5, AAV6, AAV7, AAV8, AAV9, AAV10, AAV11, AAV12, AAV13, AAV14, AAV15, AAV16, AAV.rh8, AAV.rh10, AAV.rh20, AAV.rh39, AAV.Rh74, AAV.RHM4-1, AAV.hu37, AAV.Anc80, AAV.Anc80L65, AAV.7m8, AAV.PHP.B, AAV.PHP.EB, AAV2.5, AAV2tYF, AAV3B, AAV.LK03, AAV.HSC1, AAV.HSC2, AAV.HSC3, AAV.HSC4, AAV.HSC5, AAV.HSC6, AAV.HSC7, AAV.HSC8, AAV.HSC9, AAV.HSC10, AAV.HSC11, AAV.HSC12, AAV.HSC13, AAV.HSC14, AAV.HSC15, AAV-TT, AAV-DJ8, 또는 AAV.HSC16을 제한 없이 포함하는, 여러 AAV 혈청형 중 임의의 것으로부터 유래될 수 있다. 게다가, AAV 벡터 내에 선택된 폴리뉴클레오티드 서열에 측접하는 5'와 3' ITR은 그들이 의도된 대로 기능하기만 하면, 예를 들면, 숙주 세포 유전체 또는 벡터로부터 관심되는 서열의 절제와 구조를 가능하게 하고, 그리고 AAV Rep 유전자 산물이 세포 내에 존재할 때 이종성 서열의 수용자 세포 유전체 내로의 통합을 가능하게 하기만 하면, 동일하거나 또는 동일한 AAV 혈청형 또는 단리물로부터 유래될 필요가 없다. 추가적으로, AAV ITR은 그 중에서도 특히, AAV1, AAV2, AAV3, AAV4, AAV5, AAV6, AAV7, AAV8, AAV9, AAV10, AAV11, AAV12, AAV13, AAV14, AAV15, AAV16, AAV.rh8, AAV.rh10, AAV.rh20, AAV.rh39, AAV.Rh74, AAV.RHM4-1, AAV.hu37, AAV.Anc80, AAV.Anc80L65, AAV.7m8, AAV.PHP.B, AAV.PHP.EB, AAV2.5, AAV2tYF, AAV3B, AAV.LK03, AAV.HSC1, AAV.HSC2, AAV.HSC3, AAV.HSC4, AAV.HSC5, AAV.HSC6, AAV.HSC7, AAV.HSC8, AAV.HSC9, AAV.HSC10 , AAV.HSC11, AAV.HSC12, AAV.HSC13, AAV.HSC14, AAV.HSC15, AAV-TT, AAV-DJ8, 또는 AAV.HSC16을 제한 없이 포함하는, 여러 AAV 혈청형 중에서 임의의 것으로부터 유래될 수 있다.
용어 "안티센스 올리고뉴클레오티드" 및 "ASO"는 표적 mRNA 분자 (예를 들면, Grik2 mRNA)와의 상보성 염기 대합을 통해 혼성화하고, 그리고 mRNA 불안정화 및 분해, 또는 번역의 저해를 통해 이의 발현을 저해할 수 있는 올리고뉴클레오티드를 지칭한다. ASO의 무제한적 실례는 짧은 간섭 RNA (siRNA), 짧은 헤어핀 RNA (shRNA) 및 마이크로RNA (miRNA)를 포함한다.
용어 "cDNA"는 mRNA 서열의 DNA 등가물 (다시 말하면, 우리딘이 티미딘으로 치환됨)인 핵산 서열을 지칭한다. 일반적으로, 용어 cDNA 및 mRNA는 특정 유전자 (예를 들면, Grik2 유전자)에 관하여 교체가능하게 이용될 수 있는데, 그 이유는 당업자가 우리딘이 티미딘으로서 판독된다는 것을 제외하고, cDNA 서열이 mRNA 서열과 동일하다는 것을 이해할 것이기 때문이다.
용어 "코딩 서열"은 단백질 또는 이의 일부를 인코딩하는 mRNA 분자의 핵산 서열에 상응한다. 이와 관련하여, "비코딩 서열"은 단백질 또는 이의 일부를 인코딩하지 않는 mRNA 분자의 핵산 서열에 상응한다. 비코딩 서열의 무제한적 실례는 5'와 3' 비번역 영역 (UTR), 인트론, 폴리A 꼬리, 프로모터, 인핸서, 종결자, 그리고 다른 시스-조절 서열을 포함한다.
두 번째 뉴클레오티드 또는 뉴클레오시드 서열과 관련하여 첫 번째 뉴클레오티드 또는 뉴클레오시드 서열을 설명하는 데 이용될 때 용어 "상보성"은 일정한 조건하에, 두 번째 뉴클레오티드 서열을 포함하는 올리고뉴클레오티드 또는 폴리뉴클레오티드에 혼성화하고 이것과 이중나선 구조를 형성하는, 첫 번째 뉴클레오티드 서열을 포함하는 올리고뉴클레오티드 또는 폴리뉴클레오티드의 능력을 지칭한다. 이런 조건은 예를 들면, 엄격한 조건일 수 있는데, 여기서 엄격한 조건은 다음을 포함할 수 있다: 400 mM NaCl, 40 mM PIPES pH 6.4, 1 mM EDTA, 50 ℃, 또는 70 ℃, 12-16 시간, 그 이후에 세척 (참조: 예를 들면, "Molecular Cloning: A Laboratory Manual, Sambrook, et al. (1989) Cold Spring Harbor Laboratory Press). 생물체 내부에서 목격될 수 있는 바와 같은 다른 조건, 예컨대 생리학적으로 유관한 조건이 적용될 수 있다. 혼성화된 뉴클레오티드 또는 뉴클레오시드의 궁극적 적용에 따라서 두 서열의 상보성의 검사에 가장 적합한 조건의 세트를 결정하는 방법은 당해 분야에서 널리 알려져 있다.
본원에서 이용된 바와 같이, "상보성" 서열은 또한, 혼성화하는 능력에 대한 상기 요건이 실현되는 한에 있어서, 비-왓슨 크릭 염기쌍 및/또는 비-자연 및 대안적 뉴클레오티드로부터 형성된 염기쌍을 포함하거나, 또는 이들로부터 전적으로 형성될 수 있다. 이런 비-왓슨 크릭 염기쌍은 G:U Wobble 또는 Hoogstein 염기 대합을 포함하지만 이들에 한정되지 않는다. 올리고뉴클레오티드 및 본원에서 설명된 바와 같은 표적 서열 사이의 상보성 서열은 어느 한쪽 또는 양쪽 뉴클레오티드 서열의 전장에 걸쳐, 두 번째 뉴클레오티드 서열을 포함하는 올리고뉴클레오티드 또는 폴리뉴클레오티드에 대한 첫 번째 뉴클레오티드 서열을 포함하는 올리고뉴클레오티드 또는 폴리뉴클레오티드의 염기 대합을 포함한다. 이런 서열은 본원에서 서로에 대하여 "완전히 상보성" 으로서 지칭될 수 있다. 첫 번째 서열이 본원에서 두 번째 서열에 대하여 "실제적으로 상보성" 으로서 지칭되는 경우에, 이들 두 개의 서열은 완전히 상보성일 수 있거나, 또는 이들은 그들의 궁극적 적용, 예를 들면, mRNA, 예컨대 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 데 가장 적절한 조건하에 혼성화하는 능력을 유지하면서, 혼성화 시에 30개 염기쌍까지의 이중나선에 대해 하나 이상, 그러나 일반적으로 10, 9, 8, 7, 6, 5, 4, 3, 2, 또는 1개 이내의 부정합된 염기쌍을 형성할 수 있다. 예를 들면, 폴리뉴클레오티드는 만약 상기 서열이 관심되는 mRNA의 비-중단된 부분에 실제적으로 상보적이면, 관심되는 mRNA의 적어도 일부에 상보적이다.
용어 "상보성의 영역"은 내인성 유전자 (예를 들면, Grik2)의 발현을 간섭하기 위해, 유전자, 일차 전사체, 서열 (예를 들면, 표적 서열), 또는 처리된 mRNA의 전부 또는 일부에 실제적으로 상보적인 올리고뉴클레오티드 상에서 영역을 지칭한다. 상보성의 영역이 표적 서열에 완전히 상보적이지는 않는 경우에, 분자의 내부 또는 말단 영역에 부정합이 있을 수 있다. 일반적으로, 가장 관용된 부정합은 말단 영역 내에, 예를 들면, 올리고뉴클레오티드의 5' 및/또는 3' 말단의 5, 4, 3, 또는 2개 뉴클레오티드 내에 있다.
용어 "보존성 아미노산 치환", "보존성 치환" 및 "보존성 돌연변이"는 유사한 물리화학적 특성, 예컨대 극성, 정전 전하 및 입체 용적을 나타내는 하나 이상의 상이한 아미노산에 대해 하나 이상의 아미노산의 치환을 지칭한다. 이들 특성은 아래의 표 1에서 20개의 자연 발생 아미노산 각각에 대해 요약된다.
표 1. 자연 발생 아미노산의 대표적인 물리화학적 특성
이러한 표로부터, 보존성 아미노산 패밀리는 (i) G, A, V, L 및 I; (ii) D 및 E; (iii) C, S 및 T; (iv) H, K 및 R; (v) N 및 Q; 그리고 (vi) F, Y 및 W를 포함하는 것으로 인지된다. 보존성 돌연변이 또는 치환은 이런 이유로, 하나의 아미노산을 동일한 아미노산 패밀리의 구성원에 대해 치환하는 것 (예를 들면, Thr에 대한 Ser의 치환, 또는 Arg에 대한 Lys의 치환)이다.
관용구 "세포를 올리고뉴클레오티드, 예컨대 본원에서 개시된 올리고뉴클레오티드와 접촉시키는 것"은 임의의 가능한 수단에 의해 세포를 접촉시키는 것을 포함한다. 세포를 올리고뉴클레오티드와 접촉시키는 것은 시험관내에서 세포를 올리고뉴클레오티드와 접촉시키거나 또는 생체내에서 세포를 올리고뉴클레오티드와 접촉시키는 것을 포함한다. 세포를 폴리뉴클레오티드와 접촉시키는 것은 또한, 세포를 상기 폴리뉴클레오티드를 인코딩하는 핵산 벡터 또는 이것을 내포하는 약학적 조성물과 접촉시키는 것을 지칭할 수 있다. 접촉시키는 것은 직접적으로 또는 간접적으로 행위될 수 있다. 따라서, 예를 들면, 올리고뉴클레오티드는 본원 발명의 방법을 수행하는 개체에 의해 세포와 물리적으로 접촉될 수 있거나, 또는 대안으로, 올리고뉴클레오티드 작용제는 차후에 세포와 접촉하도록 허용하거나 야기하는 상황에 놓일 수 있다. 시험관내에서 세포를 접촉시키는 것은 예를 들면, 상기 세포를 올리고뉴클레오티드와 함께 배양함으로써 행위될 수 있다. 생체내에서 세포를 접촉시키는 것은 예를 들면, 올리고뉴클레오티드 작용제가 차후에, 접촉되는 세포가 위치하는 조직에 도달하도록, 올리고뉴클레오티드를 상기 세포가 위치하는 조직 내로 또는 이와 가깝게 주사함으로써, 또는 상기 올리고뉴클레오티드 작용제를 다른 부위, 예를 들면, 혈류 또는 피하 공간 내로 주사함으로써 행위될 수 있다. 시험관내와 생체내 접촉 방법의 조합 역시 가능하다. 예를 들면, 세포는 또한, 시험관내에서 올리고뉴클레오티드와 접촉되고, 차후에 개체 내로 이식될 수 있다.
세포를 올리고뉴클레오티드와 접촉시키는 것은 세포 내로 섭취 또는 흡수를 가능하게 하거나 또는 달성함으로써, "올리고뉴클레오티드를 세포 내로 도입하는" 또는 "전달하는" 것을 포함한다. 올리고뉴클레오티드의 섭취 또는 흡수는 비보조 확산성 또는 활성 세포 과정을 통해, 또는 보조 작용제 또는 장치에 의해 발생할 수 있다. 올리고뉴클레오티드를 세포 내로 도입하는 것은 시험관내 및/또는 생체내일 수 있다. 예를 들면, 생체내 도입을 위해, 올리고뉴클레오티드(들)는 조직 부위 내로 주사되거나 또는 전신 투여될 수 있다. 세포 내로 시험관내 도입은 당해 분야에서 공지된 방법 예컨대 전기천공 및 리포펙션을 포함한다. 다른 실례에서, 올리고뉴클레오티드는 형질도입에 의해, 예컨대 폴리뉴클레오티드를 인코딩하는 바이러스 벡터에 의하여 세포 내로 도입될 수 있다. 바이러스 벡터는 인코딩된 폴리뉴클레오티드를 발현하기 위해, 세포 처리 (예를 들면, 세포 내재화, 캡시드 벗겨짐, 폴리뉴클레오티드의 전사, 그리고 Drosha 및 Dicer에 의한 처리)를 겪을 수 있다. 추가 접근방식은 하기에서 설명되고 및/또는 당해 분야에서 알려져 있다.
유전자 (예를 들면, Grik2)에 대하여 용어 "발현을 교란한다," "발현을 저해한다," 또는 "발현을 감소시킨다"는 기능적 유전자 산물 (예를 들면, GluK2 단백질)의 형성을 예방하거나 또는 감소시키는 것을 지칭한다. 유전자 산물은 만약 정상적인 (야생형) 기능(들)을 실현하면 기능적이다. 유전자의 발현의 교란은 상기 유전자에 의해 인코딩된 기능적 단백질의 발현을 예방하거나 또는 감소시킨다. 유전자 발현은 예를 들면, 간섭 RNA 분자 (예를 들면, ASO), 예컨대 본원에서 설명된 것들을 이용함으로써 교란될 수 있다.
용어 "효과량," "치료 효과량" 및 "충분한 양"의 본원에서 설명된 조성물, 벡터 작제물, 또는 바이러스 벡터는 포유동물, 예를 들면 인간을 비롯한, 개체에게 투여될 때, 임상 결과를 비롯한, 유익한 또는 원하는 결과를 달성하는 데 충분한 양을 지칭한다. 따라서, "효과량" 또는 이의 동의어는 이것이 적용되는 맥락에 좌우된다. 예를 들면, 측두엽 뇌전증 (TLE)을 치료하는 맥락에서, 이것은 조성물, 벡터 작제물, 또는 바이러스 벡터의 투여 없이 획득된 반응과 비교하여, 치료 반응을 달성하는 데 충분한 조성물, 벡터 작제물, 또는 바이러스 벡터의 양이다. 이런 양에 상응할, 본원에서 설명된 소정의 조성물의 양은 다양한 인자, 예컨대 소정의 작용제, 약학 제제, 투여 루트, 질환 또는 장애의 유형 및 이의 심각도, 개체의 정체 (예를 들면, 연령, 성별, 체중), 치료되는 숙주 및/또는 뇌전증의 경우에, 뇌전증 병소의 크기 (예를 들면, 뇌 용적) 등에 따라서 변할 것이지만, 그럼에도 불구하고 당해 분야에서 널리 알려진 방법에 따라서 결정될 수 있다. 또한, 본원에서 이용된 바와 같이, 본원 발명의 조성물, 벡터 작제물, 또는 바이러스 벡터의 "치료 효과량"은 대조와 비교하여 개체에서 유익한 또는 원하는 결과를 야기하는 양이다. 본원에서 규정된 바와 같이, 본원 발명의 조성물, 벡터 작제물, 바이러스 벡터, 또는 세포의 치료 효과량은 당해 분야에서 공지된 방법, 예컨대 본원에서 설명된 이들 방법에 의해 쉽게 결정될 수 있다. 투약 섭생은 적합한 종결점 치료 반응 (예를 들면, 치료를 받은 개체에서 뇌전증 발작의 발생에서 통계학적으로 유의한 감소)을 제공하기 위해 조정될 수 있다.
용어 "뇌전증"은 재발성 뇌전증 발작을 임상적으로 나타내는 한 가지 이상의 신경 질환을 지칭한다. 뇌전증은 Classification and Terminology of the International League Against Epilepsy (ILAE; Berg et al., 2010)에 따른 임상 전기 증후군에 따라서 분류될 수 있다. 이들 증후군은 발병 시점에 연령, 특유한 무리 (외과적 증후군), 그리고 구조-물질대사 원인에 의해 분류될 수 있다, 예컨대: (A) 발병 시점에 연령: (i) 신생아기는 양성 가족성 신생아 뇌전증 (BFNE), 초기 근간대성 뇌병증 (EME), 오타하라 증후군을 포함하고; (ii) 영아기는 이주성 초점성 발작을 동반한 영아기의 뇌전증, 웨스트 증후군, 영아기의 근간대 뇌전증 (MEI), 양성 영아 뇌전증, 양성 가족성 영아 뇌전증, 드라베 증후군, 비진행성 장애에서 근간대성 뇌병증을 포함하고; (iii) 아동기는 열성 발작 플러스 (FS+), 파나이오토풀로스 증후군, 근간대성 무긴장 (이전에 기립불능) 발작을 동반한 뇌전증, 중심 측두엽 극파를 동반한 양성 뇌전증 (BECTS), 상염색체 우성 야간 전두엽 뇌전증 (ADNFLE), 후발성 아동기 후두 뇌전증 (가스타우트형), 근간대성 소발작을 동반한 뇌전증, 레녹스 가스타우트 증후군, 수면 중 지속적인 극서파 (CSWS)를 동반한 뇌전증성 뇌병증, 랜도 크레프너 증후군 (LKS), 아동기 소발작 뇌전증 (CAE)을 포함하고; (iv) 청소년기 - 성인기는 청소년기 소발작 뇌전증 (JAE), 청소년기 근간대 뇌전증 (JME), 전신 강직성 간대 발작 단독을 동반한 뇌전증, 진행성 근간대경련 뇌전증 (PME), 청각 특징을 동반한 상염색체 우성 뇌전증 (ADEAF), 다른 가족성 측두엽 뇌전증을 포함하고; (v) 발병 연령이 가변적 것들은 가변적 병소를 동반한 가족성 초점성 뇌전증 (아동기 내지 성인기), 반사 뇌전증을 포함하고; (B) 특유한 무리 (외과적 증후군)는 내측 측두엽 뇌전증 (MTLE), 라스무센 증후군, 시상하부 과오종을 동반한 젤라스틱 발작, 반경련-반마비-뇌전증을 포함하고; (C) 구조-물질대사 원인에 기인하고 이들에 의해 조직화된 뇌전증은 피질 발달의 기형 (편측거대뇌증, 헤테로토피아 등), 신경피부 증후군 (결절성 경화증 복합체 및 스티치 웨버), 종양, 감염, 외상, 혈관종, 출생전후 손상 및 뇌졸중을 포함한다. 용어 "난치성 뇌전증"은 약학적 치료에 불응성인 뇌전증을 지칭한다; 다시 말하면 현재의 약학적 치료로는 환자의 질환의 효과적인 치료가 가능하지 않다 (참조: 예를 들면 Englot et al. J Neurosurg Pediatr 12:134-41 (2013)).
용어 "엑손"은 유전자 (예를 들면, Grik2 유전자)의 코딩 영역 내에 영역을 지칭하는데, 이것의 뉴클레오티드 서열은 상응하는 단백질의 아미노산 서열을 결정한다. 용어 "엑손"은 또한, 유전자로부터 전사된 RNA의 상응하는 영역을 지칭한다. 엑손은 프리-mRNA로 전사되고, 그리고 유전자의 대안적 스플라이싱에 따라서 성숙 mRNA 내에 포함될 수 있다. 처리 이후에 성숙 mRNA 내에 포함되는 엑손은 단백질로 번역된다. 엑손의 서열은 단백질의 아미노산 조성을 결정한다. 대안으로, 성숙 mRNA 내에 포함되는 엑손은 비코딩 (예를 들면, 단백질로 번역되지 않는 엑손)일 수 있다.
유전자 또는 핵산의 발현의 맥락에서 이용될 때 용어 "발현"은 유전자에 내포된 정보의 유전자 산물로의 전환을 지칭한다. 유전자 산물은 유전자의 직접적인 전사 산물 (예를 들면, mRNA, tRNA, rRNA, 안티센스 RNA, 리보자임, 구조 RNA 또는 임의의 다른 유형의 RNA) 또는 mRNA의 번역에 의해 생산된 단백질일 수 있다. 유전자 산물은 또한, 캡핑, 폴리아데닐화, 메틸화 및 편집과 같은 과정에 의해 변형되는 mRNA, 그리고 예를 들면, 메틸화, 아세틸화, 인산화, 유비퀴틴화, 수모화, ADP-리보실화, 미리스토일화 및 글리코실화에 의해 변형된 단백질 (예를 들면, GluK2)을 포함한다.
용어 "발현한다"는 하기 사건 중에서 한 가지 이상을 지칭한다: (1) DNA 서열로부터 RNA 주형의 생산 (예를 들면, 전사에 의해); (2) RNA 전사체의 처리 (예를 들면, 스플라이싱, 편집, 5' 캡 형성 및/또는 3' 단부 처리에 의해); (3) RNA의 폴리펩티드 또는 단백질로의 번역; 그리고 (4) 폴리펩티드 또는 단백질의 번역후 변형. 개체에서 관심되는 유전자의 발현은 예를 들면, 개체로부터 획득된 샘플 내에서 상응하는 단백질을 인코딩하는 mRNA의 양 또는 농도에서 감소 또는 증가 (예를 들면, 본원에서 설명되거나 또는 당해 분야에서 공지된 RNA 검출 절차, 예컨대 정량적 중합효소 연쇄 반응 (qPCR) 및 RNA seq 기술을 이용하여 사정될 때), 상응하는 단백질의 양 또는 농도에서 감소 또는 증가 (예를 들면, 본원에서 설명되거나 또는 당해 분야에서 공지된 단백질 검출 방법, 예컨대 그 중에서도 특히, 효소 결합 면역흡착 검정 (ELISA)을 이용하여 사정될 때), 및/또는 상응하는 단백질의 활성에서 감소 또는 증가 (예를 들면, 이온 통로의 경우에, 본원에서 설명되거나 또는 당해 분야에서 공지된 전기생리학적 방법을 이용하여 사정될 때)를 검출함으로써 현성할 수 있다.
"GluR6", "GRIK2", "MRT6", "EAA4" 또는 "GluK6"으로서 또한 알려져 있는 용어 "GluK2"는 현재 이용되는 IUPHAR 명명법에서 명명된 바와 같은 글루타민산염 이온성 수용체 카이네이트 유형 아단위 2 단백질을 지칭한다 (Collingridge, G.L., Olsen, R.W., Peters, J., Spedding, M., 2009. A nomenclature for ligand-gated ion channels. Neuropharmacology 56, 2-5). 용어 "GluK2 내포 KAR," "GluK2 수용체," "GluK2 단백질" 및 "GluK2 아단위"는 본 명세서 전반에서 교체가능하게 이용될 수 있고, 그리고 일반적으로, Grik2 유전자에 의해 인코딩되거나 또는 발현되는 단백질을 지칭한다.
용어, "가이드 가닥" 또는 "가이드 서열"은 스템 루프 구조의 5' 또는 3' 스템 루프 팔 중 어느 하나 상에 배치된 스템 루프 RNA 구조 (예를 들면, shRNA 또는 마이크로RNA)의 구성요소를 지칭하는데, 여기서 가이드 가닥/서열은 Grik2 mRNA에 결합하고 이것의 발현을 저해할 수 있는 Grik2 mRNA 안티센스 서열 (예를 들면, 서열 번호: 1-100 중에서 하나, 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)을 포함한다. 가이드 가닥/서열은 또한, 추가의 서열, 예컨대 예를 들면, 스페이서 또는 링커 서열을 포함할 수 있다. 가이드 서열은 스템 루프 RNA 구조의 승객 가닥/서열에 상보적 또는 실제적으로 상보적 (예를 들면, 5, 4, 3, 2, 또는 1개 이내의 부정합을 가짐)일 수 있다.
용어 "이온성 글루타민산염 수용체"는 NMDA (N-메틸-D-아스파르트산염), AMPA (α-아미노-3-히드록시-5-메틸-4-이속사졸프로프리온산) 및 카이네이트 수용체 (KAR) 부류의 구성원을 포함한다. 기능적 KAR은 GluK1, GluK2, GluK3, GluK4 및 GluK5 아단위로 명명된 5개 아단위의 동종체 또는 이종체 조합으로부터 사합체성 조립체로 조립될 수 있다 (Reiner et al., 2012). 본원 발명의 표적은 일부 경우에, GluK2 및 GluK5로 구성되는 KAR 복합체이다. GluK5 아단위만으로는 기능적 동종체 통로를 형성하지 않는다는 관찰 결과를 감안할 때, Grik2 유전자의 발현을 저해하는 것은 GluK2/GluK5 카이네이트 수용체 기능을 전폐하는 데 충분하다.
"발현의 저해제"는 유전자, 예를 들면, Grik2 유전자의 발현을 저해하거나 또는 감소시키는 생물학적 효과를 갖는 작용제 (예를 들면, 본원 발명의 ASO 작용제)를 지칭한다. 유전자, 예를 들면, Grik2 유전자의 발현을 저해하는 것은 비록 다양한 수준의 저해가 달성될 수 있긴 하지만, 표적 세포 또는 조직에서 유전자 산물 (단백질, 예를 들면, GluK2 단백질)의 감소 또는 심지어 전폐를 전형적으로 야기할 것이다. 발현을 저해하거나 또는 감소시키는 것은 전형적으로 녹다운으로서 지칭된다.
용어 "단리된 폴리뉴클레오티드"는 두 개 이상의 공유적으로 연결된 뉴클레오티드를 포함하는 단리된 분자를 지칭한다. 이런 공유적으로 연결된 뉴클레오티드는 핵산 분자로서 또한 지칭될 수 있다. 일반적으로, "단리된" 폴리뉴클레오티드는 자신이 획득되는 핵산 서열에 대하여 인공, 화학적으로 합성된, 정제된 및/또는 이종성인 폴리뉴클레오티드를 지칭한다.
용어 "마이크로RNA"는 mRNA 번역을 조절하여 표적 단백질 존재비에 영향을 주는 비코딩 RNA의 짧은 (예를 들면, 전형적으로 ~22개 뉴클레오티드) 서열을 지칭한다. 일부 마이크로RNA는 단일, 단일시스트론성 유전자로부터 전사되고, 반면 다른 것들은 다중유전자 유전자 클러스터의 일부로서 전사된다. 마이크로RNA의 구조는 5'와 3' 측부 서열, 스템 및 스템 루프 서열을 포함하는 헤어핀 서열을 포함할 수 있다. 세포 내에 처리 동안, 미성숙 마이크로RNA는 5'와 3' 측부 서열을 개열하는 Drosha에 의해 절두된다. 차후에, 마이크로RNA 분자는 핵에서 세포질로 전좌되는데, 여기서 이것은 Dicer에 의한 루프 영역의 개열을 겪는다. 마이크로RNA의 생물학적 작용은 mRNA 분자의 영역, 전형적으로 3' 비번역 영역에 결합을 통해 번역 조절의 수준에서 발휘되고, 그리고 mRNA의 개열, 분해, 불안정화 및/또는 덜 효율적인 번역을 야기한다. 표적에 대한 마이크로RNA의 결합은 일반적으로, 마이크로RNA의 헤어핀 서열 내에 짧은 (예를 들면, 6-8개 뉴클레오티드) "시드 영역"에 의해 매개된다. 본 명세서 전반에서, 용어 siRNA는 miRNA가 그의 등가 siRNA로서 표적 (예를 들면, 시드 영역에서)에 대해 상동성을 갖는 동일한 염기를 포괄하도록, 그의 등가 miRNA를 포함할 수 있다. 본원에서 설명된 바와 같이, 마이크로RNA는 비-자연 발생 마이크로RNA, 예컨대 하나 이상의 이종성 핵산 서열을 갖는 마이크로RNA일 수 있다.
용어 "뉴클레오티드"는 변형된 또는 자연 발생 데옥시리보뉴클레오티드 또는 리보뉴클레오티드로서 규정된다. 뉴클레오티드는 전형적으로, 티미딘, 시티딘, 구아노신, 아데노신 및 우리딘을 포함하는 퓨린과 피리미딘을 포함한다. 본원에서 이용된 바와 같이, 용어 "올리고뉴클레오티드"는 앞서 규정된 뉴클레오티드 또는 본원에서 개시된 변형된 뉴클레오티드의 소중합체로서 규정된다. 용어 "올리고뉴클레오티드"는 단일 가닥 또는 이중 가닥일 수 있는, 3'-5' 또는 5'-3' 배향된 핵산 서열을 지칭한다. 본원 발명의 맥락에서 이용되는 올리고뉴클레오티드는 특히, DNA 또는 RNA일 수 있다. 상기 용어는 또한, (i) 변형된 백본 구조, 예를 들면, 자연 올리고뉴클레오티드 및 폴리뉴클레오티드에서 발견되는 표준 포스포디에스테르 연쇄 이외의 백본 및 (ii) 임의적으로, 리보오스 또는 데옥시리보오스 모이어티가 아닌 변형된 당 모이어티, 예를 들면, 모르폴리노 모이어티를 갖는 올리고뉴클레오티드를 지칭하는 "올리고뉴클레오티드 유사체"를 포함한다. 올리고뉴클레오티드 유사체는 왓슨 크릭 염기 대합에 의해 표준 폴리뉴클레오티드 염기에 수소 결합할 수 있는 염기를 뒷받침하는데, 여기서 유사체 백본은 올리고뉴클레오티드 유사체 분자 및 표준 폴리뉴클레오티드 {예를 들면, 단일 가닥 RNA 또는 단일 가닥 DNA}에서 염기 사이에 서열 특이적 방식으로, 이런 수소 결합을 허용하는 방식으로 이들 염기를 제공한다. 특히, 유사체는 실제적으로 하전되지 않은, 인 내포 백본을 갖는 것들이다. 올리고뉴클레오티드 유사체에서 실제적으로 하전되지 않은, 인 내포 백본은 대다수, 예를 들면, 50-100% 사이의 아단위 연쇄, 전형적으로 적어도 60% 내지 100% 또는 75% 또는 80%의 연쇄가 하전되지 않고 단일 아인산 원자를 내포하는 것이다. 게다가, 용어 "올리고뉴클레오티드"는 전사를 위한 정상적인 방향에 비하여 반전되고, 따라서 숙주 세포 내에서 발현된 표적 유전자 mRNA 분자에 상보적인 RNA 또는 DNA 서열에 상응하는 올리고뉴클레오티드 서열을 지칭한다. 안티센스 가이드 가닥은 이것이 표적 유전자의 발현을 간섭할 수 있다면, 다수의 상이한 방식으로 작제될 수 있다. 예를 들면, 안티센스 가이드 가닥은 그의 보체의 전사를 가능하게 하기 위해 전사를 위한 정상적인 방향에 비하여 표적 유전자의 코딩 영역 (또는 이의 부분)을 역보체화함으로써 작제될 수 있다 (예를 들면, 안티센스 및 센스 유전자에 의해 인코딩된 RNA는 상보적일 수 있다). 올리고뉴클레오티드는 표적 유전자와 동일한 인트론 또는 엑손 패턴을 가질 필요가 없고, 그리고 표적 유전자의 비코딩 분절은 표적 유전자 발현의 안티센스 억제를 달성하는 데, 코딩 분절 예컨대 ASO와 동등하게 효과적일 수 있다. 일부 경우에, ASO는 표적 유전자와 동일한 엑손 패턴을 갖는다.
올리고뉴클레오티드는 Grik2 mRNA에 표적화 및 혼성화를 허용하는 임의의 길이일 수 있고 (예를 들면, 올리고뉴클레오티드는 적어도 Grik2 mRNA의 영역에 완벽하게 또는 실제적으로 상보적이고), 그리고 길이에서 약 10-50개 염기쌍, 예를 들면, 길이에서 약 15-50개 염기쌍 또는 길이에서 약 18-50개 염기쌍의 범위, 예를 들면, 길이에서 약 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 또는 50개 염기쌍, 예컨대 길이에서 약 15-30, 15-29, 15-28, 15-27, 15-26, 15-25, 15-24, 15-23, 15-22, 15-21, 15-20, 15-19, 15-18, 15-17, 18-30, 18-29, 18-28, 18-27, 18-26, 18-25, 18-24, 18-23, 18-22, 18-21, 18-20, 19-30, 19-29, 19-28, 19-27, 19-26, 19-25, 19-24, 19-23, 19-22, 19-21, 19-20, 20-30, 20-29, 20-28, 20-27, 20-26, 20-25, 20-24,20-23, 20-22, 20-21, 21-30, 21-29, 21-28, 21-27, 21-26, 21-25, 21-24, 21-23, 또는 21-22개 염기쌍의 범위 안에 있을 수 있다. 상기 언급된 범위와 길이의 중간 범위와 길이 또한, 본원 발명의 일부인 것으로 예기된다.
용어 "승객 가닥" 및 "승객 서열"은 Grik2 mRNA 안티센스 서열 (예를 들면, 서열 번호: 1-100 중에서 하나 또는 서열 번호: 1-108 중에서 하나의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 상보적인 또는 실제적으로 상보적인 (예를 들면, 5, 4, 3, 2, 또는 1개 이내의 부정합을 갖는) 서열을 포함하는 스템 루프 구조의 5' 또는 3' 스템 루프 팔 중 어느 하나 상에 배치된 스템 루프 RNA 구조 (예를 들면, shRNA 또는 마이크로RNA)의 구성요소를 지칭한다. 승객 가닥/서열은 또한, 추가 서열, 예컨대 예를 들면, 스페이서 또는 링커 서열을 포함할 수 있다. 승객 서열은 스템 루프 RNA 구조의 가이드 가닥/서열에 상보적이거나 또는 실질적으로 상보적일 수 있다.
용어 "플라스미드"는 추가의 DNA 분절이 결찰될 수 있는 염색체외 환상 이중 가닥 DNA 분자를 지칭한다. 플라스미드는 자신에게 연결된 다른 핵산을 수송할 수 있는 핵산 분자인 벡터의 한 가지 유형이다. 일정한 플라스미드는 그들이 도입되는 숙주 세포에서 자동 복제될 수 있다 (예를 들면, 세균 복제 기점을 갖는 세균 플라스미드 및 에피솜 포유류 플라스미드). 다른 벡터 (예를 들면, 비에피솜 포유류 벡터)는 숙주 세포 내로 도입 시에 숙주 세포의 유전체 내로 통합되고, 그것에 의하여 숙주 유전체와 함께 복제될 수 있다. 일정한 플라스미드는 그들이 작동가능하게 연결되는 유전자의 발현을 주도할 수 있다.
폴리뉴클레오티드 (예를 들면, Grik2 mRNA) 내에 개별 뉴클레오티드에 적용될 때, 용어 "위치 엔트로피"는 mRNA 이차 구조에 의해 부과된 제약 및 국부 위상을 감안할 때, 뉴클레오티드가 취할 수 있는 분자 위치, 입체형상, 또는 배열의 수를 나타내는 열역학적 양을 지칭한다. 특정한 뉴클레오티드 위치에서 낮은 위치 엔트로피는 이러한 뉴클레오티드가 적은 숫자의 위치 입체형상을 점유할 수 있다는 것을 암시한다. 특정한 뉴클레오티드 위치에서 높은 위치 엔트로피는 뉴클레오티드가 많은 수의 위치 입체형상을 점유할 수 있다는 것을 암시한다. 폴리뉴클레오티드 사슬 내에 뉴클레오티드는 다른 뉴클레오티드와의 염기 대합에 관련되고, 그것에 의하여 염기 대합된 뉴클레오티드가 취할 수 있는 위치 입체형상의 총수를 제약하는 결과로서, 낮은 위치 엔트로피를 나타낼 수 있다. 역으로, 폴리뉴클레오티드 내에 뉴클레오티드는 비혼성화되고, 그것에 의하여 염기 대합된 뉴클레오티드에 비하여 위치 입체형상에 대하여 더 많은 자유도를 갖는 결과로서, 높은 위치 엔트로피를 나타낼 수 있다. 용어 "평균 위치 엔트로피"는 소정의 서열의 모든 뉴클레오티드 위치의 전역에서 위치 엔트로피 값의 평균값을 지칭한다. 예를 들면, 평균 위치 엔트로피는 적어도 2개 (예를 들면, 적어도 2, 5, 10, 15, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 80, 85, 90, 95, 100, 125, 150, 175, 200, 300, 400, 500, 600, 700, 800, 900, 1000개, 또는 그 초과)의 뉴클레오티드에 걸쳐 계산될 수 있다. 특정 실례에서, 평균 위치 엔트로피는 2개 이상의 뉴클레오티드에 걸쳐 계산된다. 다른 실례에서, 평균 위치 엔트로피는 5개 이상의 뉴클레오티드에 걸쳐 계산된다. 다른 실례에서, 평균 위치 엔트로피는 10개 이상의 뉴클레오티드에 걸쳐 계산된다. 다른 실례에서, 평균 위치 엔트로피는 15개 이상의 뉴클레오티드에 걸쳐 계산된다. 다른 실례에서, 평균 위치 엔트로피는 20개 이상의 뉴클레오티드에 걸쳐 계산된다. 다른 실례에서, 평균 위치 엔트로피는 25개 이상의 뉴클레오티드에 걸쳐 계산된다. 다른 실례에서, 평균 위치 엔트로피는 30개 이상의 뉴클레오티드에 걸쳐 계산된다. 다른 실례에서, 평균 위치 엔트로피는 35개 이상의 뉴클레오티드에 걸쳐 계산된다. 다른 실례에서, 평균 위치 엔트로피는 40개 이상의 뉴클레오티드에 걸쳐 계산된다. 다른 실례에서, 평균 위치 엔트로피는 45개 이상의 뉴클레오티드에 걸쳐 계산된다. 다른 실례에서, 평균 위치 엔트로피는 50개 이상의 뉴클레오티드에 걸쳐 계산된다. 다른 실례에서, 평균 위치 엔트로피는 55개 이상의 뉴클레오티드에 걸쳐 계산된다. 다른 실례에서, 평균 위치 엔트로피는 60개 이상의 뉴클레오티드에 걸쳐 계산된다. 다른 실례에서, 평균 위치 엔트로피는 65개 이상의 뉴클레오티드에 걸쳐 계산된다. 다른 실례에서, 평균 위치 엔트로피는 70개 이상의 뉴클레오티드에 걸쳐 계산된다. 다른 실례에서, 평균 위치 엔트로피는 75개 이상의 뉴클레오티드에 걸쳐 계산된다. 다른 실례에서, 평균 위치 엔트로피는 80개 이상의 뉴클레오티드에 걸쳐 계산된다. 다른 실례에서, 평균 위치 엔트로피는 85개 이상의 뉴클레오티드에 걸쳐 계산된다. 다른 실례에서, 평균 위치 엔트로피는 90개 이상의 뉴클레오티드에 걸쳐 계산된다. 다른 실례에서, 평균 위치 엔트로피는 95개 이상의 뉴클레오티드에 걸쳐 계산된다. 다른 실례에서, 평균 위치 엔트로피는 100개 이상의 뉴클레오티드에 걸쳐 계산된다.
폴리뉴클레오티드 서열 내에 뉴클레오티드의 위치 엔트로피를 정량하는 방법은 당해 분야에서 널리 알려져 있다. 접힘 알고리즘 (예컨대 RNAfold)에서 예측한 바와 같이, 높은 위치 엔트로피 (제로에 더 가까움; kcal/mol)를 갖는 단일 가닥 폴리뉴클레오티드, 예컨대 mRNA 또는 RNA 저해제의 이차 구조는 자체 구조, 예컨대 스템 루프 내에 강한, 안정된 이중나선을 형성할 낮은 가능성을 갖는다. 이러한 예측된 높은 위치 엔트로피, 단일 가닥 RNA는 전형적으로, 결합 표적에 대해 높은 친화성을 나타낸다 (예를 들면, 2015년 5월 21일 자 공개된 PCT 국제 공개 번호 WO2015/073360을 참조한다). Grik2 mRNA의 비대합 영역 (비대합된 루프 및 비대합된 스템)은 높은 위치 엔트로피 (제로에 더 가까운 값; kcal/mol)를 가질 것으로 예측되고 가이드 서열과의 상호작용에 대해 호의적이다.
용어 "프로모터"는 RNA 중합효소에 의해 결합되는, DNA 상에서 인식 부위를 지칭한다. 중합효소는 폴리뉴클레오티드의 전사를 주동한다. 본원에서 설명된 조성물과 방법용으로 적합한 예시적인 프로모터는 예를 들면 Sandelin et al., Nature Reviews Genetics 8:424 (2007)에서 설명되는데, 이것의 개시는 핵산 조절 요소와 관련되는 한에 있어서, 본원에서 참조로서 편입된다. 추가적으로, 용어 "프로모터"는 생물학적 시스템에서 자연적으로 발생하지 않는 조절 DNA 서열인 합성 프로모터를 지칭할 수 있다. 합성 프로모터는 자연에서 발생하지 않고 다양한 폴리뉴클레오티드, 벡터 및 표적 세포 유형을 이용하여 재조합 DNA를 발현하도록 최적화될 수 있는 폴리뉴클레오티드 서열과 조합된 자연 발생 프로모터의 일부를 내포한다.
참조 폴리뉴클레오티드 또는 폴리펩티드 서열에 대하여 "서열 동일성 퍼센트 (%)"는 최대 서열 동일성 퍼센트를 달성하기 위해 서열을 정렬하고 필요하면, 갭을 도입한 후, 참조 폴리뉴클레오티드 또는 폴리펩티드 서열 내에 핵산 또는 아미노산과 동일한 후보 서열 내에 핵산 또는 아미노산의 백분율로서 규정된다. 핵산 또는 아미노산 서열 동일성 퍼센트를 결정하는 목적으로 정렬은 당해 분야에서 널리 알려져 있는 다양한 방식으로, 예를 들면, 공개적으로 가용한 컴퓨터 소프트웨어 예컨대 BLAST, BLAST-2, 또는 Megalign 소프트웨어를 이용하여 달성될 수 있다. 비교되는 서열의 전장에 걸쳐 최대 정렬을 달성하는 데 필요한 임의의 알고리즘을 비롯한, 충분히 인정된 전통적인 방법을 이용하여, 서열을 정렬하기 위한 적합한 파라미터가 결정될 수 있다. 예를 들면, 서열 동일성 퍼센트 값은 서열 비교 컴퓨터 프로그램 BLAST를 이용하여 생성될 수 있다. 예시로서, 소정의 핵산 또는 아미노산 서열, B에 대한 소정의 핵산 또는 아미노산 서열, A (대안으로 소정의 핵산 또는 아미노산 서열, B에 대해 일정한 서열 동일성 퍼센트를 갖는 소정의 핵산 또는 아미노산 서열, A로서 표현될 수 있다)의 서열 동일성 퍼센트는 아래와 같이 계산되고:
100 곱하기 (분율 X/Y)
여기서 X는 서열 정렬 프로그램 (예를 들면, BLAST)의 A 및 B의 정렬에서 상기 프로그램에 의해 동일한 정합으로서 채점된 뉴클레오티드 또는 아미노산의 수이고, 그리고 여기서 Y는 B에서 핵산의 총수이다. 핵산 또는 아미노산 서열 A의 길이가 핵산 또는 아미노산 서열 B의 길이와 동등하지 않는 경우에, B에 대한 A의 서열 동일성 퍼센트는 A에 대한 B의 서열 동일성 퍼센트와 동등하지 않을 것으로 인지될 것이다.
용어 "약학적으로 허용되는"은 합리적인 유익성/위험 비율에 비례하여 과도한 독성, 자극, 알레르기 반응 및 다른 문제 합병증 없이 개체, 예컨대 포유동물 (예를 들면, 인간)의 조직과의 접촉에 적합한 이들 화합물, 물질, 조성물 및/또는 약형을 지칭한다.
본원에서 이용된 바와 같이, 용어 "약학적 조성물"은 약학적으로 허용되는 부형제로 조제된 본원에서 설명된 화합물 (예를 들면, ASO 또는 이것을 내포하는 벡터)을 내포하는 조성물을 나타내고, 그리고 일부 경우에 포유동물에서 질환의 치료를 위한 치료 섭생의 일부로서 정부 규제 당국의 승인하에 제조되거나 또는 판매될 수 있다. 약학적 조성물은 예를 들면, 단위 약형 (예를 들면, 정제, 캡슐, 당의정, 젤라틴 캡슐, 또는 시럽)에서 경구 투여용으로, 국소 투여용으로 (예를 들면, 크림, 겔, 로션, 또는 연고로서), 정맥내 투여용으로 (예를 들면, 미립자 색전이 없는 무균 용액으로서 및 정맥내 이용에 적합한 용매 시스템에서), 척수강내 주사용으로, 뇌실내 주사용으로, 뇌실질내 주사용으로, 또는 임의의 다른 약학적으로 허용되는 제제에서 조제될 수 있다.
"약학적으로 허용되는 부형제"는 환자에서 실제적으로 비독성 및 비염증성의 특성을 갖는, 본원에서 설명된 화합물 이외에 임의의 성분 (예를 들면, 활성 화합물을 현탁하거나 또는 용해할 수 있는 운반제)을 지칭한다. 부형제는 예를 들면: 부착방지제, 항산화제, 결합제, 코팅, 압축 보조제, 붕괴제, 염료 (컬러), 피부연화제, 유화제, 충전제 (희석제), 도막 형성제 또는 코팅, 풍미제, 향기, 활택제 (흐름 개선제), 윤활제, 보존제, 인쇄용 잉크, 흡수제, 현탁제 또는 분산제, 감미료, 그리고 수화수를 포함할 수 있다. 예시적인 부형제는 부틸화된 히드록시톨루엔 (BHT), 탄산칼슘, 인산칼슘 (이염기성), 스테아르산칼슘, 크로스카르멜로스, 교차연결된 폴리비닐 피롤리돈, 구연산, 크로스포비돈, 시스테인, 에틸셀룰로오스, 젤라틴, 히드록시프로필 셀룰로오스, 히드록시프로필 메틸셀룰로오스, 락토오스, 스테아르산마그네슘, 말티톨, 만니톨, 메티오닌, 메틸셀룰로오스, 메틸 파라벤, 미정질 셀룰로오스, 폴리에틸렌 글리콜, 폴리비닐 피롤리돈, 포비돈, 전호화된 전분, 프로필 파라벤, 레티닐 팔미트산염, 셸락, 이산화실리콘, 나트륨 카르복시메틸 셀룰로오스, 구연산나트륨, 나트륨 전분 글리콜산염, 소르비톨, 전분 (옥수수), 스테아르산, 수크로오스, 활석, 이산화티타늄, 비타민 A, 비타민 E, 비타민 C, 그리고 자일리톨을 포함하지만 이들에 한정되지 않는다.
본원에서 설명된 화합물 (예를 들면, ASO 및 이들을 내포하는 벡터)은 약학적으로 허용되는 염으로서 제조할 수 있기 위해 이온화가능 기를 가질 수 있다. 이들 염은 무기 또는 유기 산을 수반하는 산 부가염일 수 있거나 또는 이들 염은 본원에서 설명된 화합물의 산성 형태의 경우에, 무기 또는 유기 염기로부터 제조될 수 있다. 빈번하게는, 화합물은 약학적으로 허용되는 산 또는 염기의 부가물로서 제조된 약학적으로 허용되는 염으로서 제조되거나 또는 이용된다. 적합한 약학적으로 허용되는 산과 염기 및 적합한 염의 제조 방법은 당해 분야에서 널리 알려져 있다. 염은 무기와 유기 산과 염기를 비롯한 약학적으로 허용되는 비독성 산과 염기로부터 제조될 수 있다. 대표적인 산 부가염은 아세트산염, 아디핀산염, 알긴산염, 아스코르브산염, 아스파르트산염, 벤젠술폰산염, 벤조산염, 중황산염, 붕산염, 부티르산염, 캄포산염, 캄포술폰산염, 구연산염, 시클로펜탄프로피온산염, 디글루코네이트, 도데실술페이트, 에탄술폰산염, 푸마르산염, 글루코헵토네이트, 글리세로포스페이트, 헤미술페이트, 헵토네이트, 헥사노에이트, 브롬화수소산염, 염산염, 요오드화수소산염, 2-히드록시-에탄술폰산염, 락토비오네이트, 유산염, 라우린산염, 라우릴 황산염, 말산염, 말레인산염, 말론산염, 메탄술폰산염, 2-나프탈렌술폰산염, 니코틴산염, 질산염, 올레산염, 옥살산염, 팔미트산염, 파모산염, 펙틴산염, 과황산염, 3-페닐프로피온산염, 인산염, 피크르산염, 피발레이트, 프로피온산염, 스테아르산염, 숙신산염, 황산염, 주석산염, 티오시안산염, 톨루엔술폰산염, 운데카노에이트 및 발레르산염 염을 포함한다. 대표적인 알칼리 또는 알칼리성 토류 금속 염은 나트륨, 리튬, 칼륨, 칼슘 및 마그네슘뿐만 아니라, 암모늄, 테트라메틸암모늄, 테트라에틸암모늄, 메틸아민, 디메틸아민, 트리메틸아민, 트리에틸아민 및 에틸아민을 포함하지만 이들에 한정되지 않는 비독성 암모늄, 사차 암모늄 및 아민 양이온을 포함한다.
용어 "조절 서열"은 유전자의 전사 또는 번역을 제어하는 프로모터, 인핸서 및 다른 발현 제어 요소 (예를 들면, 폴리아데닐화 신호)를 포함한다. 이런 조절 서열은 예를 들면, 본원에서 참조로서 편입되는 Perdew et al., Regulation of Gene Expression (Humana Press, New York, NY, (2014))에서 설명된다.
용어 "표적" 또는 "표적화"는 상보성 염기 대합을 통해, GluK2 단백질을 인코딩하는 Grik2 유전자 또는 mRNA에 특이적으로 결합하는 ASO 작용제 (예를 들면, 예컨대 본원에서 설명된 ASO 작용제)의 능력을 지칭한다.
용어 "단일 가닥 영역"은 단일 가닥 (예를 들면, mRNA 내에 다른 뉴클레오티드에 비혼성화됨)이거나 또는 실제적으로 단일 가닥 (예를 들면, 동일한 Grik2 mRNA 분자의 다른 뉴클레오티드에 혼성화된 영역 내에 5% 이내의 뉴클레오티드를 가짐)인 Grik2 mRNA (예를 들면, 서열 번호: 115의 핵산 서열 또는 서열 번호: 115의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 Grik2 mRNA)의 예측된 이차 구조의 영역에 상응한다. Grik2 mRNA의 단일 가닥 영역의 무제한적 실례는 Grik2 mRNA (서열 번호: 115)의 예측된 루프 영역 1-14 (서열 번호: 145-158) 및 예측된 비대합 영역 1-5 (서열 번호: 159-163)를 포함하였다.
용어 "짧은 간섭 RNA" 및 "siRNA"는 각 가닥이 RNA, RNA 유사체(들) 또는 RNA와 DNA를 포함하는 이중 가닥 핵산을 지칭한다. siRNA 분자는 19개 및 23개 사이의 뉴클레오티드 (예를 들면, 21개의 뉴클레오티드)를 포함할 수 있다. siRNA는 siRNA 내에 이중나선 영역이 17-21개의 뉴클레오티드 (예를 들면, 19개의 뉴클레오티드)를 포함하도록, 각 가닥의 3' 단부 상에서 전형적으로 2 bp 오버행을 갖는다. 전형적으로, siRNA의 안티센스 가닥은 표적 유전자/RNA의 표적 서열과 충분히 상보적이다. siRNA 분자는 RNA 간섭 경로 내에 작동하여, 표적 mRNA (예를 들면, Grik2 mRNA)에 결합하고 mRNA를 Dicer 매개 mRNA 개열을 통해 분해함으로써 mRNA 발현의 저해를 야기한다. 본 명세서 전반에서, 용어 siRNA는 miRNA가 그의 등가 siRNA로서 표적에 대해 상동성을 갖는 동일한 염기를 포괄하도록, 그의 등가 miRNA를 포함하는 것으로 의미된다.
용어 "짧은 헤어핀 RNA" 및 "shRNA"는 세포에서 스템 루프 구조를 형성하는 50 내지 100개 뉴클레오티드의 단일 가닥 RNA를 지칭하는데, 이것은 5 내지 30개 뉴클레오티드의 루프 영역, 그리고 상기 루프 영역의 양쪽 측면에 15 내지 50개 뉴클레오티드의 긴 상보성 RNA를 내포하고, 이들은 상보성 RNA 서열 사이의 염기 대합에 의해 이중 가닥 스템을 형성하며; 그리고 일부 경우에, 스템을 형성하는 각 상보성 가닥 전후에 추가의 1 내지 500개 뉴클레오티드가 포함된다. 예를 들면, shRNA는 일반적으로, RNA 중합효소에 의해 전사를 종결하기 위해 헤어핀의 3'에 특정한 서열을 필요로 한다. 이런 shRNA는 일반적으로, 짧은 5'와 3' 측부 서열의 포함으로 인해, Drosha에 의한 처리를 우회한다. 다른 shRNA, 예컨대 RNA 중합효소 II로부터 전사되는 "shRNA 유사 마이크로RNA"는 더 긴 5'와 3' 측부 서열을 포함하고, 그리고 핵에서 Drosha에 의한 처리를 필요로 하며, 그 후 개열된 shRNA가 핵에서 시토졸로 이출되고 시토졸에서 Dicer에 의해 더욱 개열된다. siRNA와 마찬가지로, shRNA는 표적 mRNA에 서열 특이적 방식으로 결합하고, 그것에 의하여 표적 mRNA를 개열하고 파괴하여, 표적 mRNA의 발현을 억제한다.
용어 "개체" 및 "환자"는 동물 (예를 들면, 포유동물, 예컨대 인간)을 지칭한다. 본원에서 설명된 방법에 따라서 치료를 받는 개체는 뇌전증 (예를 들면, TLE)으로 진단된 개체, 또는 이러한 질환이 발달할 위험에 처해 있는 개체일 수 있다. 진단은 당해 분야에서 공지된 임의의 방법 또는 기술에 의해 수행될 수 있다. 본원 발명에 따른 치료를 받는 개체는 표준 검사가 진행될 수 있거나, 또는 조사 없이, 질환 또는 장애와 연관된 하나 이상의 위험 요인의 존재로 인해 위험에 처해 있는 개체로서 확인될 수 있었다.
용어 "측두엽 뇌전증" 또는 "TLE"은 뇌의 측두엽에서 유래하는 만성 재발성 발작 (뇌전증)에 의해 특징화되는 만성 신경학적 장애를 지칭한다. 이 질환은 미경험 뇌 조직에서 급성 발작과 상이한데, 그 이유는 TLE는 뉴런 네트워크의 형태 기능적 재편성 및 해마의 치상회의 과립 세포로부터 재발성 이끼 섬유의 발아에 의해 특징화되는 반면, 미경험 조직에서 급성 발작은 이런 회로 특이적 재편성을 촉발시키지 않기 때문이다. TLE는 뇌의 어느 한쪽 또는 양쪽 반구에서 뇌전증유발 병소의 출현으로부터 발생할 수 있다.
용어 "형질도입" 및 "형질도입한다"는 핵산 물질 (예를 들면, 벡터, 예컨대 바이러스 벡터 작제물, 또는 이의 일부)을 세포 내로 도입하고, 그리고 차후에, 세포에서 핵산 물질 (예를 들면, 상기 벡터 작제물 또는 이의 일부)에 의해 인코딩된 폴리뉴클레오티드를 발현하는 방법을 지칭한다.
용어 "치료" 또는 "치료한다"는 질환에 걸릴 위험에 처해 있거나 또는 질환에 걸린 것으로 의심되는 환자뿐만 아니라 병들었거나 또는 질환 또는 의학적 상태를 겪는 것으로 진단된 환자의 치료를 비롯한, 방지적 및 예방적 처치 둘 모두뿐만 아니라 치유력 있는 또는 질환 변경 치료를 지칭한다. 치료는 또한, 임상적 재발의 억제를 포함한다. 치료는 장애 또는 재발성 장애를 예방하거나, 이것을 치유하거나, 이것의 개시를 지연시키거나, 이것의 심각도를 감소시키거나, 또는 이것의 한 가지 이상의 증상을 개선하기 위해, 또는 이런 치료의 부재 시에 예상된 것을 뛰어넘어 개체의 생존을 연장하기 위해 의학적 장애를 겪고 있거나 또는 이러한 장애에 궁극적으로 걸릴 수 있는 개체에게 시행될 수 있다. "치료 섭생"은 질병의 치료의 패턴, 예를 들면, 요법 동안 이용된 투약의 패턴인 것으로 의미된다. 치료 섭생은 유도 섭생 및 유지 섭생을 포함할 수 있다. 관용구 "유도 섭생" 또는 "유도기"는 질환의 초기 치료에 이용되는 치료 섭생 (또는 치료 섭생의 일부)을 지칭한다. 유도 섭생의 일반적인 목적은 치료 섭생의 초기 기간 동안 높은 수준의 약물을 환자에게 제공하는 것이다. 유도 섭생은 "부하 섭생"을 이용할 수 있는데 (부분적으로 또는 전체적으로), 이것은 유지 섭생 동안 의사가 이용할 용량보다 더 큰 용량의 약물을 투여하는 단계, 유지 섭생 동안 의사가 약물을 투여할 빈도보다 더 빈번하게 약물을 투여하는 단계, 또는 둘 모두를 포함할 수 있다. 관용구 "유지 섭생" 또는 "유지기"는 예를 들면, 환자를 긴 기간 (수개월 또는 수년) 동안 관해 상태로 유지하기 위해 질병의 치료 동안 환자의 유지에 이용되는 치료 섭생 (또는 치료 섭생의 일부)을 지칭한다. 유지 섭생은 연속 요법 (예를 들면, 약물을 규칙적인 간격으로, 예를 들면, 주 1회, 월 1회, 연 1회 등으로 투여), 또는 간헐적 요법 (예를 들면, 치료 중단, 간헐적 치료, 재발 시에 치료, 또는 특정한 미리 결정된 기준 (예를 들면, 질환 현성)의 달성 시에 치료)을 이용할 수 있다.
용어 "벡터"는 핵산 벡터, 예를 들면, DNA 벡터, 예컨대 플라스미드, RNA 벡터, 또는 다른 적합한 레플리콘 (예를 들면, 바이러스 벡터)을 포함한다. 외인성 폴리뉴클레오티드 또는 단백질을 인코딩하는 폴리뉴클레오티드의 원핵 또는 진핵 세포로의 전달을 위한 다양한 벡터가 개발되었다. 이런 발현 벡터의 실례는 예를 들면, WO 1994/011026에서 개시되는데, 이것은 관심되는 핵산 물질의 발현에 적합한 벡터와 관련되는 한에 있어서, 본원에서 참조로서 편입된다. 본원에서 설명된 조성물과 방법용으로 적합한 발현 벡터는 폴리뉴클레오티드 서열뿐만 아니라, 예를 들면, 포유류 세포에서 이종성 핵산 물질 (예를 들면, ASO)의 발현에 이용되는 추가의 서열 요소를 내포한다. 본원에서 설명된 ASO 작용제의 발현에 이용될 수 있는 일정한 벡터는 유전자 전사를 주도하는 조절 서열, 예컨대 프로모터와 인핸서 영역을 내포하는 플라스미드를 포함한다. 본원에서 개시된 ASO 작용제의 발현을 위한 다른 유용한 벡터는 이들 폴리뉴클레오티드의 번역 속도를 증강하거나 또는 유전자 전사로부터 발생하는 RNA의 안정성 또는 핵 이출을 향상시키는 폴리뉴클레오티드 서열을 내포한다. 이들 서열 요소는 예를 들면, 발현 벡터 상에서 운반되는 유전자의 효율적인 전사를 주도하기 위한 5'와 3' 비번역 영역, IRES 및 폴리아데닐화 신호 부위를 포함한다. 본원에서 설명된 조성물과 방법용으로 적합한 발현 벡터는 또한, 이런 벡터를 내포하는 세포의 선택을 위한 마커를 인코딩하는 폴리뉴클레오티드를 내포할 수 있다. 적합한 마커의 실례는 항생제, 예컨대 암피실린, 클로람페니콜, 카나마이신, 누르세오트리신, 또는 제오신에 대한 내성을 인코딩하는 유전자이다.
용어 "결합의 총 자유 에너지"는 mRNA 상에서 표적 영역 개방, 단일 가닥 가이드의 생성, 그리고 단일 가닥 siRNA 가이드의 그의 단일 가닥 mRNA 표적 서열에 혼성화를 포함하는, Grik2 mRNA (예를 들면, 서열 번호: 115) 상에서 상응하는 표적 서열에 혼성화하는 뉴클레오티드 또는 폴리뉴클레오티드 (예를 들면, 본원 발명의 ASO 작용제)의 과정의 자유 에너지에 상응하는 뉴클레오티드 또는 폴리뉴클레오티드의 열역학적 특성 (kcal/mol로 계측됨)을 지칭한다. 본원 발명의 맥락에서, 특정 ASO 서열에 대한 결합의 총 자유 에너지의 더 음성 값은 상기 ASO에 의한 Grik2 mRNA 발현의 녹다운의 감소된 효능과 일반적으로 연관되고, 반면 제로에 더 가까운 값은 일반적으로 증가된 녹다운 효능을 반영한다.
용어 "이중나선 형성으로부터 에너지"는 단일 가닥 mRNA 서열 (예를 들면, Grik2 mRNA)에 대한 단일 가닥 siRNA 가이드의 혼성화의 자유 에너지에 상응하는 뉴클레오티드 또는 폴리뉴클레오티드의 열역학적 특성 (kcal/mol로 계측됨)을 지칭한다. 본원 발명의 맥락에서, 소정의 뉴클레오티드 또는 폴리뉴클레오티드에 대한 이중나선 형성의 더 음성 에너지 값은 이중나선의 형성이, 이중나선 형성의 에너지가 제로에 더 가까운 이중나선의 형성보다 더 호의적이라는 것을 반영하고, 그리고 또한 Grik2 mRNA 발현의 감소된 녹다운 효능을 반영한다. 이런 이유로, 이러한 값은 이중나선 형성의 호감도 및 녹다운 효능 사이의 역 관계를 표시하고, 이중나선 형성의 에너지가 이중나선 형성보다는 이중나선 분리 (이의 역)의 호감도를 결정하기 위한 더 강한 척도를 제공한다는 것을 암시한다. 따라서, 이중나선 형성으로부터 에너지의 값이 더 음성일수록, 이중나선이 더 안정된다. 덜 안정된 Grik2 표적:ASO 이중나선은, ASO가 아마도 증가된 진행도로 인해 Grik2 mRNA 발현을 녹다운시키는 데 더 유효할 가능성이 높다는 것을 표시할 수 있다는 결론이 나온다. 다시 말하면, 표적 서열과 복합된 ASO는 상이한 mRNA 분자 상에서 동일한 영역을 표적으로 하기 위해 덜 안정된 이중나선에서 떨어져 나올 가능성이 더 높은데, 이것은 그의 녹다운 효능을 반영할 것이다.
용어 "개방 에너지" 및 "총 개방 에너지"는 표적 위치에서 RNA 이차 구조를 분해하는 (다시 말하면, 개방하는/접근 가능하게 만드는) 데 필요한 에너지에 상응하고, 그리고 인근 이차 구조의 분해 또는 표적 서열과 이차 구조를 형성하는 원위 서열의 관여를 잠재적으로 포함하는 뉴클레오티드 또는 폴리뉴클레오티드의 열역학적 특성 (kcal/mol로 계측됨)을 지칭한다. 본원 발명의 맥락에서, 개방 에너지의 더 음성 값은 RNA 이차 구조를 분해하기 위한 더 높은 에너지 요구량을 표시하고 상응하는 ASO 서열의 감소된 녹다운 효능을 반영한다. 이러한 값은 중첩풀림에 더 적은 에너지를 필요로 하는 표적 서열이 중첩풀림에 더 순응하고, 그리고 이런 이유로 ASO 결합에 대해 접근성이 더 높은 것으로 고려될 수 있다는 것을 암시한다.
용어 "GC 함량" 및 "퍼센트 (%) GC"는 구아닌 (G) 또는 시토신 (C) 중 어느 하나인, 폴리뉴클레오티드 (예를 들면, 본원 발명의 ASO 또는 이의 완전히 또는 실제적으로 상보성 서열) 내에 염기의 백분율을 지칭한다. 2개의 수소 결합에 의해 매개되는 A-T/U 결합과 달리, G-C 결합은 3개의 수소 결합에 의해 매개된다. 더 많은 GC 함량을 갖는 폴리뉴클레오티드 이중나선은 더 안정되고 이중나선을 분해하기 위해 더 많은 에너지를 필요로 한다. 이러한 안정성은 증가된 수의 수소 결합에 의해 반드시 부여되기 보다는, 더 안정된 염기 스태킹에 의해 부여된다. 소정의 폴리뉴클레오티드에 대해, GC 함량은 아래와 같이 계산될 수 있다:
도면의 간단한 설명
도 1a-1q는 세포에서 GluK2 단백질의 발현을 녹다운 (KD)시키기 위한 글루타민산염 이온성 수용체 카이네이트 유형 아단위 2 (Grik2) mRNA 안티센스 올리고뉴클레오티드 (ASO) 작제물의 확인과 사정을 보여준다. (도 1a) 중심 엔트로피 모형인 RNAfold에 의해 예측된 바와 같은 인간 Grik2 mRNA 변이체 1 (서열 번호: 115)의 예측된 이차 구조. (도 1b) Grik2 mRNA에 대한 복수의 항-Grik2 ASO의 결합의 예측된 영역. 결합의 예시적인 영역은 확인된 루프 영역 (1 = 루프 1 (서열 번호: 145); 2 = 루프 2 (서열 번호: 146); 3 = 루프 3 (서열 번호: 147); 4 = 루프 4 (서열 번호: 148); 5 = 루프 5 (서열 번호: 149); 6 = 루프 6 (서열 번호: 150); 7 = 비대합된 1 (서열 번호: 159); 8 = 비대합된 2 (서열 번호: 160); 9 = 루프 7 (서열 번호: 151); 10 = 루프 8 (서열 번호: 152); 11 = 루프 9 (서열 번호: 153); 12 = 루프 10 (서열 번호: 154); 13 = 비대합된 3 (서열 번호: 161); 14 = 루프 11 (서열 번호: 155); 15 = 비대합된 4 (서열 번호: 162); 16 = 비대합된 5 (서열 번호: 163); 17 = 루프 12 (서열 번호: 156); 18 = 루프 13 (서열 번호: 157); 및 19 = 루프 14 (서열 번호: 158)), 그리고 스템 유사 비대합 영역 (각각 서열 번호: 159-163에 상응하는 아라비아 숫자 15-19에 의해 지정됨)을 포함한다. (도 1c) Grik2 mRNA (서열 번호: 115)의 5' UTR 내에 결합의 예측된 영역에 맞춰 정렬된 항-Grik2 ASO 작용제 (서열 번호: 126; siRNA D3 (서열 번호: 48), siRNA XZ (서열 번호: 54), siRNA CY (서열 번호: 43), siRNA D1 (서열 번호: 46), siRNA GE (서열 번호: 65), siRNA CX (서열 번호: 42), siRNA Y0 (서열 번호: 55), siRNA TG (서열 번호: 23), siRNA D0 (서열 번호: 45), siRNA YB (서열 번호: 67), siRNA GF (서열 번호: 64), siRNA TD (서열 번호: 26), siRNA GH (서열 번호: 66), siRNA TE (서열 번호: 25), siRNA TJ (서열 번호: 21), siRNA TF (서열 번호: 24), siRNA YB/siSPOTR15 (서열 번호: 67), siRNA ZZ/siSPOTR16 (서열 번호: 100), siRNA GE/siSPOTR17 (서열 번호: 65), siRNA D3/siSPOTR18 (서열 번호: 48), siRNA CX/siSPOTR19 (서열 번호: 42), siRNA GF/siSPOTR20 (서열 번호: 64), siRNA GH/siSPOTR21 (서열 번호: 66), siRNA TJ/siSPOTR22 (서열 번호: 21), siRNA TG/siSPOTR23 (서열 번호: 23), siRNA TD/siSPOTR24 (서열 번호: 26) 및 siRNA TF/siSPOTR25 (서열 번호: 24)), 그리고 엑손 1의 코딩 서열 (CDS) (서열 번호: 129; siRNA CK (서열 번호: 29), siRNA TC (서열 번호: 28) 및 siRNA TC/siSPOTR1 (서열 번호: 28))의 계통도. (도 1d) Grik2 mRNA (서열 번호: 115)의 엑손 2 (서열 번호: 130) 내에 확인된 루프 1 영역 (서열 번호: 145)에 상대적으로 정렬된 예시적인 ASO 작용제 (G0; 서열 번호: 1)의 계통도. (도 1e) Grik2 mRNA (서열 번호: 115)의 엑손 10 (서열 번호: 138) 내에 확인된 루프 5 (서열 번호: 149) 및 루프 6 (서열 번호: 150) 영역에 상대적으로 정렬된 5개의 예시적인 ASO 서열 (GD (서열 번호: 7), MU (서열 번호: 96), MT (SEQ ID NO:99), MS (서열 번호: 99) 및 G3 (서열 번호: 8))의 계통도. (도 1f) Grik2 mRNA (서열 번호: 115)의 엑손 11 (서열 번호: 139)에 상대적으로 정렬된 예시적인 ASO 작용제 (MJ (서열 번호: 89), TH (서열 번호: 22), MI (서열 번호: 90), Y9 (서열 번호: 88), TK (서열 번호: 74), Y8 (서열 번호: 87), TI (서열 번호: 76), CU (서열 번호: 39) 및 Y7 (서열 번호: 62))를 보여주는 계통도. (도 1g) 이중-루시페라아제 리포터 검정에서 다양한 후보 ASO 작용제에 의한 Grik2 mRNA의 리포터 녹다운 퍼센트 (표 2를 또한 참조한다). (도 1h) 이중-루시페라아제 리포터 검정에서 다양한 후보 항-Grik2 ASO 작용제에 대한 비특이적 반딧불이 루시페라아제 (ffluc) 감소. (도 1i) 빈 대조 벡터 (관계 표적화된 비특이적 ffluc 녹다운)로부터 잔류 ffluc 발현의 함수로서 Grik2 mRNA 녹다운 퍼센트를 보여주는 산점도. (도 1j) 루시페라아제 리포터 검정에서 검사된 19 bp siRNA 모두에 대한 표적 개방 에너지 (kcal/mol)의 평균 (SEM 포함) 값의 막대 플롯. 데이터 막대 (중간 및 오른쪽 막대)는 리포터 유발 Gluk2 발현을 66% 초과하여 녹다운시킨 siRNA, 또는 발현을 66% 미만으로 녹다운시킨 siRNA에 의해 분리되었다. 10 kcal/mol 미만의 표적 개방 에너지를 갖는 siRNA의 강화된 클러스터는 66% 초과의 Gluk2 녹다운과 상관되었다. (도 1k) 루시페라아제 리포터 검정에서 검사된 바와 같은 19 bp 등가물의 녹다운 퍼센트에 대하여 21 bp 가이드 모두에 대한 표적 개방 에너지 (kcal/mol)의 평균 (SEM 포함) 값의 막대 플롯. 데이터 막대 (중간 및 오른쪽 막대)는 리포터 유발 Gluk2 발현을 66% 초과하여 녹다운시킨 등가 siRNA, 또는 발현을 66% 미만으로 녹다운시킨 등가 siRNA에 의해 분리되었다. 9.5 kcal/mol 미만의 표적 개방 에너지를 갖는 siRNA의 강화된 클러스터는 66% 초과 Gluk2 녹다운과 상관되었다. (도 1l) 루시페라아제 리포터 검정에서 검사된 19 bp siRNA 모두의 이중나선 형성의 에너지 (kcal/mol)의 평균 (SEM 포함)의 막대 플롯. 데이터 막대는 왼쪽에서 오른쪽으로: 검사된 19 bp siRNA 모두, 리포터 발현을 >66% 녹다운시킨 siRNA, 또는 리포터 발현을 <66% 녹다운시킨 siRNA를 나타낸다. (도 1m) 루시페라아제 리포터 검정에서 검사된 바와 같은 19 bp 등가물의 녹다운 퍼센트에 대하여 21 bp 가이드 모두에 대한 이중나선 형성의 에너지 (kcal/mol)의 평균 (SEM 포함) 값의 막대 플롯. 데이터 막대 (중간 및 오른쪽 막대)는 리포터 유발 Gluk2 발현을 66% 초과하여 녹다운시킨 등가 siRNA, 또는 발현을 66% 미만으로 녹다운시킨 등가 siRNA에 의해 분리되었다. (도 1n) 루시페라아제 리포터 검정에서 검사된 19 bp siRNA 모두에 대한 총 결합 에너지 (kcal/mol)의 평균 (SEM 포함)의 막대 플롯. 데이터 막대는 왼쪽에서 오른쪽으로: 검사된 19 bp siRNA 모두, 리포터 발현을 >66% 녹다운시킨 siRNA, 또는 리포터 발현을 <66% 녹다운시킨 siRNA를 나타낸다. (도 1o) 루시페라아제 리포터 검정에서 검사된 바와 같은 19 bp 등가물의 녹다운 퍼센트에 대하여 21 bp 가이드 모두에 대한 총 결합 에너지 (kcal/mol)의 평균 (SEM 포함) 값의 막대 플롯. 데이터 막대 (오른쪽, 중간)는 리포터 유발 Gluk2 발현을 66% 초과하여 녹다운시킨 등가 siRNA, 또는 발현을 66% 미만으로 녹다운시킨 등가 siRNA에 의해 분리되었다. (도 1p) 루시페라아제 리포터 검정에서 검사된 19 bp siRNA 각각에서 G 또는 C로서 확인된 염기의 퍼센트 (GC 함량)의 평균 (SEM 포함)의 막대 플롯. 데이터 막대는 왼쪽에서 오른쪽으로: 검사된 19 bp siRNA 모두, 리포터 발현을 >66% 녹다운시킨 siRNA, 또는 리포터 발현을 <66% 녹다운시킨 siRNA를 나타낸다. (도 1q) 루시페라아제 리포터 검정에서 검사된 바와 같은 19 bp 등가물의 녹다운 퍼센트에 대하여 21 bp 가이드 모두에 대한 GC 함량의 평균 (SEM 포함) 값의 막대 플롯. 데이터 막대 (오른쪽, 중간)는 리포터 유발 Gluk2 발현을 66% 초과하여 녹다운시킨 등가 siRNA, 또는 발현을 66% 미만으로 녹다운시킨 등가 siRNA에 의해 분리되었다.
도 2a-2j는 바이러스 벡터 매개 Grik2 mRNA 침묵에 의한 GluK2 녹다운의 검증을 보여준다. (도 2a-2d) 도 2a-2j에서 설명된 실험에서 활용된 예시적인 벡터. (도 2a) hSyn 프로모터 (서열 번호: 682)의 제어하에 대조 스크램블 서열 (서열 번호: 771)을 인코딩하는 렌티바이러스 벡터 (CM845)에 대한 예시적인 렌티바이러스 플라스미드 지도. (도 2b) U6 프로모터 (서열 번호: 772)의 제어하에 shRNA로서 Grik2 안티센스 서열 (G9; 서열 번호: 68)을 인코딩하는 렌티바이러스 벡터 (CM946)에 대한 예시적인 렌티바이러스 플라스미드 지도. (도 2c) hSyn 프로모터 (서열 번호: 683)의 제어하에 miRNA로서 Grik2 안티센스 서열 (G9; 서열 번호: 68)을 인코딩하는 렌티바이러스 벡터 (CM962)에 대한 예시적인 렌티바이러스 플라스미드 지도. (도 2d) hSyn 프로모터의 제어하에 GFP를 인코딩하는 AAV 벡터 (pAAV-hSyn-EGFP; 서열 번호: 682)에 대한 예시적인 플라스미드 지도. (도 2e) 렌티바이러스 (LV)-인간 시냅신 프로모터 (hSyn (서열 번호: 682))-녹색 형광 단백질 (GFP) 플라스미드 작제물로 감염된 배양된 쥐 해마 뉴런의 Brightfield (왼쪽 패널) 및 형광 (오른쪽 패널) 영상화. GFP 면역형광이 배양된 뉴런 중 >80%에서 관찰되었다. (도 2f) Grik2 안티센스 서열 (G9; 서열 번호: 68) 또는 대조 서열 (서열 번호: 771; hSyn 프로모터 (서열 번호: 682)의 제어하에)을 인코딩하는 짧은-헤어핀 RNA (LV-U6 (서열 번호: 772)-G9 (shRNA) 또는 마이크로RNA (LV-hSyn (서열 번호: 683)-G9 (miRNA) 작제물 중 어느 하나로 Grik2 mRNA의 렌티바이러스 매개 녹다운 이후에 쥐 해마 뉴런으로부터 획득된 GluK2 단백질 및 액틴의 웨스턴 블롯. (도 2g) 세포에서 AAV 매개 바이러스 형질도입에 이용된 AAV 발현 카세트의 계통도. (도 2h) 항-Grik2 ASO 서열 (G9; 서열 번호: 68) 또는 스크램블 대조 서열 (LV: 서열 번호: 771; AAV: GC - 서열 번호: 101)을 인코딩하는 렌티바이러스 또는 AAV9 벡터로 감염된 해마 뉴런에 대한 대조 조건에서 값에 대해 정규화된, GluK2 단백질 대 액틴의 상대적 수준을 나타내는 막대 그래프. (도 2i) 상이한 바이러스로 인코딩된 항-Grik2 ASO 서열 (G9 (서열 번호: 68); GI (서열 번호: 77); XY (서열 번호: 83); Y9 (서열 번호: 88); GG (서열 번호: 91)) 및 대조 서열 (GC; 서열 번호: 101)로 처리된 뮤린 일차 피질 뉴런에서, 웨스턴 블롯에 의해 검정될 때, GluK2 단백질의 상대적 수준을 보여주는 플롯. (도 2j) RT-qPCR에 의해 계측될 때, hSyn 프로모터 (서열 번호: 683)의 조절 제어하에 5개의 Grik2 mRNA 안티센스 올리고뉴클레오티드 (G9 (서열 번호: 68), GI (서열 번호: 77), Y9 (서열 번호: 88), XY (서열 번호: 83) 또는 MU (서열 번호: 96)) 중에서 하나, 또는 스크램블 대조 서열 (GC; 서열 번호: 101)을 인코딩하는 플라스미드 벡터와 함께 17,500개 세포/웰 (17.5k c/w)의 세포 밀도로 배양된 유도 만능 줄기 세포 (iPSC) 유래된 글루타민산성 뉴런 (GlutaNeurons)의 지질 기반 형질감염 5 일 후에 계측된 Grik2 mRNA 발현에서 배수 변화의 막대 플롯을 보여준다.
도 3a-3d는 뮤린 시험관내 모형에서 해마 뇌전증모양 활성에 대한 바이러스로 인코딩된 항-Grik2 ASO 작용제의 효과를 보여준다. (도 3a) 스크램블 서열 (서열 번호: 101)을 내포하는 AAV9-hSyn (서열 번호: 682)-GFP-스크램블 작제물로 감염된 기관형적 해마 뇌 슬라이스를 보여주는 형광 이미지. 상기 슬라이스는 해마의 치상회 (DG) 세포를 표지화하기 위해 프로스페로 호메오복스 단백질 1 (Prox1) 항체 (Millipore)로 면역염색되었다. (도 3b) 뮤린 기관형적 해마 슬라이스로부터 기록된 ED의 예시적인 세포외 전압 트레이스. (도 3c) hSyn 프로모터 (LV 및 AAV9- GC: 서열 번호: 682; AAV9-hSyn-G9: 서열 번호: 683), 또는 AAV9-GFP 대조 벡터의 제어하에 miRNA 작제물로서 항-Grik2 ASO 서열 (G9; 서열 번호: 68; ***, p< 0.001; **, p<0.01) 또는 스크램블 대조 서열 (AAV- GC (서열 번호: 101); LV-스크램블 (서열 번호: 771))을 인코딩하는 렌티바이러스 또는 AAV9 벡터로 감염된 뮤린 해마 슬라이스에서 ED의 주파수를 나타내는 막대 그래프. (도 3d) 표시된 AAV9-인코딩된 항-Grik2 ASO 작용제 (AAV9-hSyn (서열 번호: 683)-G9 (서열 번호: 68) - p = 0.0004; AAV9-hSyn (서열 번호: 683)-XY (서열 번호: 91) - p = 0.0008; AAV9-hSyn (서열 번호: 683)-GI (서열 번호: 85) - p = 0.0478); AAV9-hSyn (서열 번호: 683)-Y9 (서열 번호: 96); 및 AAV9-hSyn (서열 번호: 683)-GG (서열 번호: 91) 또는 대조 스크램블 서열 및 GFP 태그 (AAV9-hSyn (서열 번호: 682)-GFP-GC (서열 번호: 101))로 처리된 뮤린 기관형적 해마 슬라이스에서 ED의 주파수를 나타내는 막대 그래프.
도 4a-4f는 측두엽 뇌전증 (TLE)의 생체내 뮤린 모형에서 Grik2 표적화 ASO 작용제의 효능을 보여준다. (도 4a) 새로운 물체 인식 (NOR) 과제의 실험 설계에 대한 계통도. (도 4b) 바이러스로 인코딩된 스크램블 서열 (GC; 서열 번호: 101; AAV9-hSyn (서열 번호: 682)-GFP-GC) 또는 항-Grik2 서열 (G9; 서열 번호: 68; AAV9-hSyn (서열 번호: 683)-G9) 중 어느 하나로 주사 7 일 전에 또는 주사 15 일 후에 계측될 때, NOR 과제가 진행된 생쥐의 식별 지수 (DI)를 보여주는 막대 그래프. (도 4c) 바이러스로 인코딩된 스크램블 서열 (GC; 서열 번호: 101) 또는 항-Grik2 서열 (G9; 서열 번호: 68) 중 어느 하나로 주사 7 일 전에 또는 주사 15 일 후에 계측될 때, NOR 과제에서 생쥐에 의한 이동된 총 거리 (cm)를 보여주는 막대 그래프. (도 4d) 필로카르핀으로 처리 이후에 생쥐로부터 기록된 바와 같은, TLE의 필로카르핀 모형에서 유도된 전기사진술 발작의 예시적인 전압 트레이스. (도 4e) 바이러스로 인코딩된 스크램블 대조 서열 (GC; 서열 번호: 101; n = 3) 또는 바이러스로 인코딩된 항-Grik2 ASO 작용제 (G9; 서열 번호: 68; n = 4)로 처리된 생쥐에서 5 일에 걸쳐 누적 발작 지속 기간 (분)을 보여주는 막대 그래프. (도 4f) 바이러스로 인코딩된 스크램블 대조 서열 (GC; 서열 번호: 101; n = 5) 또는 바이러스로 인코딩된 항-Grik2 ASO 작용제 (G9; 서열 번호: 68; n = 6)로 처리된 생쥐에서 5 일에 걸쳐 발작의 누적 횟수를 보여주는 막대 그래프.
도 5는 AAV9 벡터에서 인코딩된 다양한 Grik2 mRNA 표적화 마이크로RNA 작제물의 녹다운 효능을 보여주는 막대 그래프이다. AAV9 벡터는 A-miR-30 (S1), E-miR-30 (S2), E-miR-155 (S3), E-miR-218 (S4) 및 E-miR-124 (S5)를 비롯한, 내인성 마이크로RNA로부터 5' 측부 영역, 마이크로RNA 루프 서열 및 3' 측부 영역을 내포하는 5개의 마이크로RNA 골격 중에서 하나를 통합한다. 검사된 안티센스 서열은 G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80), GU (서열 번호: 96), TO (서열 번호: 14), TK (서열 번호: 74), TH (서열 번호: 22), CQ (서열 번호: 35), XU (서열 번호: 51), XY (서열 번호: 83), Y9 (서열 번호: 88), YA (서열 번호: 63), GG (서열 번호: 91), G8 (서열 번호: 92), ME (서열 번호: 69), 그리고 MD (서열 번호: 70)이었다. 녹다운 효능은 "지질 단독" 대조에 비하여 Grik2 mRNA 중앙 배수 변화로서 표현된다. hSyn 프로모터 (서열 번호: 790)의 제어하에, miRNA 발현 플라스미드의 이러한 더 큰 패널은 유도 만능 줄기 세포 (iPSC) 유래된 글루타민산성 뉴런 (GlutaNeurons) 내로 형질감염되고, 그리고 RT-qPCR에 의해 Grik2 mRNA 수준을 감소시키는 능력에 대해 평가되었다. 비형질감염된 세포 (파선)와 비교하고, 그리고 중위 절대 편차 (MAD) = 2를 이용하여 기능적 작제물 (점선)을 확인할 때, 대다수의 작제물은 기능적인 것으로 결정되었다 (다시 말하면, 이들은 MAD 미만에서 녹다운 Grik2 mRNA를 나타낸다). 모든 검사된 작제물 중에서, GI (서열 번호: 77)-S2 (서열 번호: 798), MW (서열 번호: 80)-S4 (서열 번호: 799), MW-S5 (서열 번호: 800), 그리고 G9 (서열 번호: 68)-S5 (서열 번호: 801)가 Grik2 mRNA를 가장 높은 정도로 녹다운 (다시 말하면, 20% 이상 녹다운)시키는 것으로 밝혀졌다.
도 6a-6g는 내인성 miRNA로부터 5' 측부 영역, 마이크로RNA 루프 서열 및 3' 측부 영역을 내포하는 A-miR-30 (S1) 골격 내로 통합된 안티센스 가이드 서열을 내포하는 합성 AAV9-miRNA 작제물 입체형상의 계통도를 보여준다. 작제물 1 (도 6a)은 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 서열 (서열 번호: 790), miR-30 5' 측부 서열 (서열 번호: 752), G9의 항-Grik2 서열 (서열 번호: 68)에 실제적으로 상보적인 승객 가닥 서열, miR-30 루프 서열 (서열 번호: 758), G9의 가이드 서열 (서열 번호: 68), miR-30 3' 측부 서열 (서열 번호: 753), 토끼 베타-글로빈 (RBG) 폴리A 신호 (서열 번호: 792), 그리고 3' ITR 서열 (서열 번호: 789)을 내포하는 단일-miRNA, 단일 프로모터 작제물 (서열 번호: 775)이다. 작제물 2 (도 6b)는 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), C1ql2 프로모터 서열 (서열 번호: 791), hSyn 프로모터 서열 (서열 번호: 790), miR-30 5' 측부 서열 (서열 번호: 752), G9의 항-Grik2 서열 (서열 번호: 68)에 실제적으로 상보적인 승객 가닥 서열, miR-30 루프 서열 (서열 번호: 785), G9의 가이드 서열 (서열 번호: 68), miR-30 3' 측부 서열 (서열 번호: 753), RBG 폴리A 신호 (서열 번호: 792), 그리고 3' ITR 서열 (서열 번호: 748)을 내포하는 단일 miRNA, 이중 프로모터 작제물 (서열 번호: 777)이다. 작제물 3 (도 6c)은 hSyn 프로모터 (서열 번호: 790)에 인접한 5' 단부 (다시 말하면, 5')에서 야생형 AAV (wt)ITR 및 폴리A 서열의 하류에서 돌연변이체 ITR (mITR)을 내포하는 자가 상보성, 이중-miRNA (G9, 서열 번호: 68의 2개 사본), 단일 프로모터 작제물 (서열 번호: 779)이다. 작제물 4 (도 6d; 서열 번호: 781)는 hSyn 프로모터 (서열 번호: 790)가 mITR에 인접하고 폴리A 서열이 wtITR에 인접하다는 점을 제외하고, 작제물 3과 유사하다. 작제물 5 (서열 번호: 783) 및 작제물 6 (서열 번호: 784)은 이들 작제물이 동일한 miRNA 서열 (예를 들면, G9, 서열 번호: 68; 작제물 5)의 3개 사본 또는 상이한 miRNA 서열 (도 6e; 작제물 6; G9, GI (서열 번호: 77), MU (서열 번호: 96))의 3개 사본을 내포하도록, 프리-miR 스템 루프 구조 (5' 측부, 스템 루프 및 3' 측부)가 3회 연쇄체화된다는 점을 제외하고, 작제물 1과 유사하다. 작제물 7 (도 6f; 서열 번호: 804)은 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 서열 (서열 번호: 790), miR-30 5' 측부 서열 (서열 번호: 752), G9의 항-Grik2 서열 (서열 번호: 68)에 실제적으로 상보적인 승객 가닥 서열, miR-30 루프 서열 (서열 번호: 758), G9의 가이드 서열 (서열 번호: 68), miR-30 3' 측부 서열 (서열 번호: 753), RBG 폴리A 신호 (서열 번호: 792), 비코딩 스터퍼 서열, 그리고 3' ITR 서열 (서열 번호: 789)을 내포하는 단일-miRNA, 단일 프로모터 작제물이다. 작제물 8 (도 6g; 서열 번호: 810)은 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 서열 (서열 번호: 790), E-miR-124-3 5' 측부 서열 (서열 번호: 768), G9의 안티센스 서열 (서열 번호: 68)에 상보적인 센스 승객 가닥 서열, E-miR-124-3 루프 서열 (서열 번호: 770), G9의 안티센스 가이드 서열, E-miR-124-3 3' 측부 서열 (서열 번호: 769), RBG 폴리A 신호 (서열 번호: 792), 비코딩 스터퍼 서열, 그리고 3' ITR 서열 (서열 번호: 789)을 내포하는 단일-miRNA, 단일 프로모터 작제물이다.
도 7은 도 6a-6e에서 설명된 단일- 및 이중-miRNA 발현 작제물 (작제물 1-6)의 알칼리성 아가로즈 겔 전기이동 분석을 보여주는 사진이다. 단일 프로모터 및 단일 miRNA 카세트 (예상된 길이: 1.5 kb)를 인코딩하는 플라스미드로부터 생성된 벡터의 유전체 내용물은 단독적으로 (1.5 kb), 이중으로 (3.0 kb) 및 삼중으로 (4.5 kb) 포장된 유전체의 혼합물로 구성되는 것으로 밝혀졌다. 레인 번호는 아래의 벡터 작제물에 상응한다: 1 = 작제물 1 (서열 번호: 775); 2 = 작제물 2 (서열 번호: 777); 3 = 작제물 3 (서열 번호: 779); 4 = 작제물 4 (서열 번호: 781); 5 = 작제물 5 (서열 번호: 783); 6 = 작제물 6 (서열 번호: 784).
도 8a-8g는 AAV 벡터용으로 적합한 이중 프로모터를 갖는 AAV9 이중-miRNA 발현 작제물의 계통도를 보여준다. 도 8a는 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 (서열 번호: 790), E-miR-124-3 5' 측부 서열 (서열 번호: 768), G9의 안티센스 서열 (서열 번호: 68)에 상보적인 센스 승객 ("P") 가닥 서열, E-miR-124-3 루프 서열 (서열 번호: 770), G9의 안티센스 가이드 ("G") 서열, E-miR-124-3 3' 측부 서열 (서열 번호: 769), BGH 폴리A 서열 (서열 번호: 793), CaMKII 프로모터 서열 (서열 번호: 802), E-miR-30 5' 측부 서열 (서열 번호: 759), GI (서열 번호: 77)에 상보적인 센스 승객 가닥 서열, E-miR-30 루프 서열 (서열 번호: 761), GI의 안티센스 가이드 서열 (서열 번호: 77), E-miR-30 3' 측부 서열 (서열 번호: 760), RBG 폴리A 서열 (서열 번호: 792), 그리고 3' ITR 서열 (서열 번호: 748)을 내포하는 이중-miRNA 이중 프로모터 벡터 (DMTPV1) 발현 작제물 (서열 번호: 785)을 보여준다. 도 8b는 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 (서열 번호: 790), E-miR-124-3 5' 측부 서열 (서열 번호: 768), G9의 안티센스 서열 (서열 번호: 68)에 상보적인 센스 승객 가닥 서열, E-miR-124-3 루프 서열 (서열 번호: 770), G9의 안티센스 가이드 서열, E-miR-124-3 3' 측부 서열 (서열 번호: 769), BGH 폴리A 서열 (서열 번호: 793), CaMKII 프로모터 서열 (서열 번호: 802), E-miR-218 5' 측부 서열 (서열 번호: 765), MW (서열 번호: 80)에 상보적인 센스 승객 가닥 서열, E-miR-218 루프 서열 (서열 번호: 767), MW의 안티센스 가이드 서열 (서열 번호: 80), E-miR-218 3' 측부 서열 (서열 번호: 766), RBG 폴리A 서열 (서열 번호: 792), 그리고 3' ITR 서열 (서열 번호: 748)을 내포하는 이중 siRNA 발현 작제물 (DMTPV2, 서열 번호: 786)을 보여준다. 도 8c는 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 (서열 번호: 790), E-miR-30 5' 측부 서열 (서열 번호: 759), GI (서열 번호: 77)에 상보적인 센스 승객 가닥 서열, E-miR-30 루프 서열 (서열 번호: 761), GI의 안티센스 가이드 서열 (서열 번호: 77), E-miR-30 3' 측부 서열 (서열 번호: 760), BGH 폴리A 서열 (서열 번호: 793), CaMKII 프로모터 서열 (서열 번호: 802), E-miR-124-3 5' 측부 서열 (서열 번호: 768), G9의 안티센스 서열 (서열 번호: 68)에 상보적인 센스 승객 가닥 서열, E-miR-124-3 루프 서열 (서열 번호: 770), G9의 안티센스 가이드 서열, E-miR-124-3 3' 측부 서열 (서열 번호: 769), RBG 폴리A 서열 (서열 번호: 792), 그리고 3' ITR 서열 (서열 번호: 748)을 내포하는 이중 siRNA 발현 작제물 (DMTPV3, 서열 번호: 787)을 보여준다. 도 8d는 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 (서열 번호: 790), E-miR-30 5' 측부 서열 (서열 번호: 759), GI (서열 번호: 77)에 상보적인 센스 승객 가닥 서열, E-miR-30 루프 서열 (서열 번호: 761), GI의 안티센스 가이드 서열 (서열 번호: 77), E-miR-30 3' 측부 서열 (서열 번호: 760), BGH 폴리A 서열 (서열 번호: 793), CaMKII 프로모터 서열 (서열 번호: 802), E-miR-124-3 5' 측부 서열 (서열 번호: 768), MW의 안티센스 서열 (서열 번호: 80)에 상보적인 센스 승객 가닥 서열, E-miR-124-3 루프 서열 (서열 번호: 770), MW의 안티센스 가이드 서열 (서열 번호: 80), E-miR-124-3 3' 측부 서열 (서열 번호: 769), RBG 폴리A 서열 (서열 번호: 792), 그리고 3' ITR 서열 (서열 번호: 748)을 내포하는 이중 siRNA 발현 작제물 (DMTPV4, 서열 번호: 788)을 보여준다. 도 8e는 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 (서열 번호: 790), E-miR-30 5' 측부 서열 (서열 번호: 759), GI (서열 번호: 77)에 상보적인 센스 승객 가닥 서열, E-miR-30 루프 서열 (서열 번호: 761), GI의 안티센스 가이드 서열 (서열 번호: 77), E-miR-30 3' 측부 서열 (서열 번호: 760), BGH 폴리A 서열 (서열 번호: 793), CaMKII 프로모터 서열 (서열 번호: 802), E-miR-218 5' 측부 서열 (서열 번호: 765), MW의 안티센스 서열 (서열 번호: 80)에 상보적인 센스 승객 가닥 서열, E-miR-218 루프 서열 (서열 번호: 767), MW의 안티센스 가이드 서열 (서열 번호: 80), E-miR-218 3' 측부 서열 (서열 번호: 766), RBG 폴리A 서열 (서열 번호: 792), 그리고 3' ITR 서열 (서열 번호: 748)을 내포하는 이중 siRNA 발현 작제물 (DMTPV5)을 보여준다. 도 8f는 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 (서열 번호: 790), E-miR-30 5' 측부 서열 (서열 번호: 759), GI (서열 번호: 77)에 상보적인 센스 승객 가닥 서열, E-miR-30 루프 서열 (서열 번호: 761), GI의 안티센스 가이드 서열 (서열 번호: 77), E-miR-30 3' 측부 서열 (서열 번호: 760), E-miR-218 5' 측부 서열 (서열 번호: 765), MW의 안티센스 서열 (서열 번호: 80)에 상보적인 센스 승객 가닥 서열, E-miR-218 루프 서열 (서열 번호: 767), MW의 안티센스 가이드 서열 (서열 번호: 80), E-miR-218 3' 측부 서열 (서열 번호: 766), RBG 폴리A 서열 (서열 번호: 792), 비코딩 스터퍼 서열, 그리고 3' ITR 서열 (서열 번호: 748)을 내포하는 이중 siRNA 발현 작제물 (DMTPV6)을 보여준다. 도 8g는 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 (서열 번호: 790), E-miR-30 5' 측부 서열 (서열 번호: 759), GI (서열 번호: 77)에 상보적인 센스 승객 가닥 서열, E-miR-30 루프 서열 (서열 번호: 761), GI의 안티센스 가이드 서열 (서열 번호: 77), E-miR-30 3' 측부 서열 (서열 번호: 760), BGH 폴리A 서열 (서열 번호: 793), CaMKII 프로모터 서열 (서열 번호: 802), E-miR-124-3 5' 측부 서열 (서열 번호: 768), G9의 안티센스 가이드 서열, E-miR-124-3 루프 서열 (서열 번호: 770), G9의 안티센스 서열 (서열 번호: 68)에 상보적인 센스 승객 가닥 서열, E-miR-124-3 3' 측부 서열 (서열 번호: 769), RBG 폴리A 서열 (서열 번호: 792), 그리고 3' ITR 서열 (서열 번호: 748)을 내포하는 이중 siRNA 발현 작제물 (DMTPV7)을 보여준다.
도 9a 및 9b는 단일-siRNA 벡터 작제물 (도 9a; G9, 서열 번호: 68 - 작제물 1 (서열 번호: 775); GC, 서열 번호: 101) 및 이중-miRNA 벡터 작제물 (도 9b; DMTPV1-4; 각각, 서열 번호: 785-788)로부터 생산된 벡터의 cDNA의 알칼리성 아가로오스 겔 분석을 보여주는 사진이다. 4개의 이중-miRNA 벡터 작제물 전체에 대하여 단일 띠는 이중 발현 작제물의 벡터가 AAV9 벡터에서 단독적으로 포장된다는 것을 암시한다.
도 10은 qPCR에 의해 계측될 때, 단독적으로 또는 상이한 miRNA 서열을 내포하는 다른 단일-miRNA AAV9 작제물 (G9-S1 (서열 번호: 775), GI-S1 (서열 번호: 796), GI-S2 (서열 번호: 798), MW-S4 (서열 번호: 799), G9-S5 (서열 번호: 800) 또는 이들의 조합)과 조합으로 전달된 단일-miRNA AAV9 작제물을 이용한 GluK2 단백질 녹다운의 시험관내 효능을 나타내는 막대 그래프를 보여준다. 두 가지 상이한 항-Grik2 miRNA 서열 (둘 모두 hSyn 프로모터 (서열 번호: 790)의 제어하에)의 조합으로 형질감염된 GlutaNeurons가 단일 유형의 항-Grik2 miRNA 서열로 형질감염된 GlutaNeurons와 유사한 GluK2 단백질 녹다운을 보여주었는데, 이것은 GluK2 발현을 녹다운시키기 위한, Grik2에 대한 하나 초과의 고유한 안티센스 가이드 서열을 인코딩하는 벡터의 이용을 뒷받침하였다. 녹다운 효능은 "지질 단독" 대조군에 비하여 중앙 Grik2 mRNA 수준 배수 변화에서 배수 변화로서 계측되었다.
도 11은 hSyn 프로모터 (서열 번호: 790)의 제어하에 상이한 단일-miRNA AAV9 발현 벡터 (GC (서열 번호: 101); G9-S1 (서열 번호: 775), GI-S1 (서열 번호: 796), 또는 G9-S1 + GI-S1)의 조합으로 형질감염된 무억제성 뮤린 기관형적 해마 슬라이스에서 뇌전증모양 활성의 주파수의 막대 그래프를 보여준다. miRNA 작제물, G9-S1 및 GI-S1의 조합은 개별적으로 각 벡터와 동등한 정도의 뇌전증모양 활성 억제를 보여주었는데, 이것은 해마 회로에서 뇌전증모양 활성을 억제하기 위한, Grik2에 대한 하나 초과의 고유한 안티센스 가이드 서열의 이용을 뒷받침하였다.
도 12는 hSyn.GI (서열 번호: 77).S2 (서열 번호: 798), hSyn.MW (서열 번호: 80).S4 (서열 번호: 799), hSyn.MW.S5 (서열 번호: 800), hSyn.G9 (서열 번호: 68).S5 (서열 번호: 801), CaMKII.GI.S4, CaMKII.MW.S5, CaMKII.G9.S5, DMTPV1 (서열 번호: 785), DMTPV2 (서열 번호: 786), DMTPV3 (서열 번호: 787) 및 DMTPV4 (서열 번호: 788)를 비롯한, 여러 안티센스 작제물 중에서 하나를 이용하여 GlutaNeurons에서 Grik2의 AAV9 벡터 매개 녹아웃 이후에, qPCR에 의해 계측될 때, Grik2 mRNA의 수준을 나타내는 막대 그래프를 보여준다. mRNA 수준은 RNA를 생산하지 않는 전장 유전체를 내포하는 AAV.null 벡터로 형질도입된 GlutaNeurons와 비교되었다.
도 13a 및 13b는 필로카르핀 및 여러 단일-miRNA 벡터 작제물 (GC (서열 번호: 101), G9-S1 (서열 번호: 775), 또는 GI-S1 (서열 번호: 796)) 중에서 하나로 처리된 생쥐를 이용하여 수행된 오픈 필드 시험의 결과를 보여준다. 도 13a는 오픈 필드에서 단일 생쥐에 대한 추적된 운동의 예시적인 트레이스를 보여준다. 도 13b는 항-Grik2 miRNA 서열을 인코딩하는 AAV9 벡터로 처리된 생쥐에 의한 오픈 필드 시험에서 이동된 총 거리를 나타내는 막대 그래프를 보여준다. 비-뇌전증 생쥐 (다시 말하면, 필로카르핀으로 처리되지 않은 생쥐; n=20) 및 GC, G9-S1 또는 GI-S1 (각각, n=9, n=8, n=9)으로 처리된 만성 뇌전증 생쥐. 주사전 및 주사후 데이터는 만 휘트니 검정, *p<0.05 및 **p<0.01을 이용하여 비교되었다. G9 및 GI에서 과운동의 유의미한 감소에 주목한다.
도 14는 항-Grik2 작제물 G9-S1 (서열 번호: 775), GI-S1 (서열 번호: 796), 또는 스크램블 대조 작제물 GC (서열 번호: 101)를 인코딩하는 AAV9 벡터로 처리된 필로카르핀-처리 생쥐 (각각, n=5, n=5, n=5)에서 하루에 뇌전증 발작의 총수를 보여주는 막대 그래프이다. G9-S1 및 GI-S1 미가공 데이터는 일원 변량 분석 검정, p<0.01을 이용하여 GC와 비교되었다. G9-S1 및 GI-S1에서 발작의 억제에 주목한다.
도 15는 상이한 용량에서 항-Grik2 작제물 G9 (서열 번호: 68)를 인코딩하는 AAV9 벡터로 처리된 생쥐에 의한 오픈 필드 시험에서 이동된 총 거리를 보여주는 막대 그래프이다. 상이한 용량의 G9: G9/1, G9/10, G9/100 및 G9/1000으로 처리된 만성 뇌전증 생쥐 (각각, n=8, n=5, n=5, n=5). GC는 대조 작제물 (n=9)이다. 주사전 및 주사후 데이터는 만 휘트니 검정, *p<0.05 및 **p<0.01을 이용하여 비교되었다. G9 및 G9/10의 유사한 효과에 주목한다.
도 16은 여러 용량: G9/1, G9/10, G9/100 및 G9/1000 중 하나에서 항-Grik2 작제물 G9-S1/G9 (서열 번호: 775)를 인코딩하는 AAV9 벡터로 처리된 필로카르핀-처리 생쥐 (각각, n=6, n=4, n=2, n=2)에서 하루에 뇌전증 발작의 총수를 보여주는 막대 그래프이다. G9 및 G9/10에서는 유사한 효과가 달성되지만, G9/1000에서는 그렇지 않다는 점에 주목한다.
도 17은 여러 이중-miRNA, 이중 프로모터 작제물 (DMTPV1-4) 중에서 하나를 인코딩하는 AAV9 벡터로 처리된 필로카르핀-처리 생쥐에 의한 오픈 필드 시험에서 이동된 총 거리를 보여주는 막대 그래프이다. 비-뇌전증 생쥐 (n=20) 및 DMTPV1 (서열 번호: 785), DMTPV2 (서열 번호: 786), DMTPV3 (서열 번호: 787) 또는 DMTPV4 (서열 번호: 788)로 처리된 만성 뇌전증 생쥐 (각각, n=6, n=5, n=6 및 n=6). 주사전 및 주사후 데이터는 만 휘트니 검정, *p<0.05 및 **p<0.01을 이용하여 비교되었다. DMTPV3 및 DMTPV4에서 과운동의 유의미한 감소에 주목한다.
도 18은 필로카르핀-처리 생쥐에서 하루에 자연발생적 뇌전증 발작의 횟수와 대비하여 오픈 필드 시험에서 이동된 총 거리 (cm)를 보여주는 산점도이다. 회귀 분석은 과운동 및 발작 감수성 사이의 유의미한 상관을 증명한다 (R2 = 0.7388, p < 0.0001).
도 19는 G9 (서열 번호: 68), GI (서열 번호: 77), DMTPV1 (서열 번호: 785), DMTPV2 (서열 번호: 786), DMTPV3 (서열 번호: 787) 및 DMTPV4 (서열 번호: 788)를 비롯한 여러 항-Grik2 miRNA 서열 중에서 하나, 그리고 대조 AAV9.hSyn.GFP 벡터로 GlutaNeurons의 형질도입 이후에 Grik2 mRNA 발현을 보여주는 막대 그래프이다. 모든 검사된 이중-miRNA 작제물은 RNA 염기서열분석을 이용하여 계측될 때, GlutaNeurons에서 Grik2 mRNA 수준을 감소시켰다. 배수 변화는 대조에 상대적이다.
도 20은 단일 항-Grik2 miRNA 서열 G9 (서열 번호: 68)를 인코딩하는 벡터, 이중-miRNA 벡터 DMTPV3 (서열 번호: 787)으로 처리된 생쥐, 또는 대조 AAV9.hSyn.GFP 벡터로 처리된 생쥐의 오픈 필드 시험에서 운동을 보여주는 막대 그래프이다. WT 생쥐는 별개의 대조로서 이용되었다. 프리 = 처리전; 포스트 = 처리후. G9- 및 DMTPV3-인코딩 벡터 둘 모두 생쥐에서 과운동 활성을 유의미하게 억제하였는데 (p < 0.01; 만 휘트니 검정), 이것은 과운동의 억제에 대한 이들 벡터의 견실한 효과를 암시하였다.
도 21은 DMTPV3 (3.6x1010개 GC/뇌), DMTPV3/10 (3.6x109개 GC/뇌), DMTPV3/100 (3.6x108개 GC/뇌) 및 DMTPV3/1000 (3.6x107개 GC/뇌)을 비롯한, 변하는 용량의 DMTPV3 (서열 번호: 787)으로 처리된 생쥐의 오픈 필드 시험에서 과운동 활성의 용량 의존성 감소를 보여주는 막대 그래프이다. 대조 생쥐는 AAV9.hSyn.GFP 벡터 (3.6x1010개 GC/뇌)로 처리되었다. 프리 = 처리전; 포스트 = 처리후. DMTPV3 및 DMTPV3/10 투약으로 처리된 생쥐는 대조 생쥐에 비하여 과운동 활성에서 유의미한 감소를 보여주었다 (p < 0.01; 만 휘트니 검정).
도 22는 항-Grik2 단일-miRNA 작제물 G9 (서열 번호: 68) 또는 GI (서열 번호: 77), 또는 이중-miRNA 작제물 DMTPV3 (서열 번호: 787)을 인코딩하는 벡터로 더욱 처리된 필로카르핀-처리 생쥐에서 하루에 발작의 횟수를 보여주는 막대 그래프이다. 생쥐는 또한, 스크램블 RNA 서열 GC (서열 번호: 101) 또는 AAV9.hSyn.GFP를 인코딩하는 대조 벡터로 처리되었다. G9, GI 및 DMTPV3으로 처리된 생쥐는 하루에 발작의 횟수에서 유의미한 감소를 보여주었는데, DMTPV가 G9 및 GI와 비교하여 더 큰 감소를 보여주었다 (* p < 0.05; ** p < 0.01; 만 휘트니 검정).
도 23은 DIV 1 이후에 AAV9.GC(SEQ 식별 번호: 101).GFP로 감염되고 치상 과립 세포의 마커 (PROX1)에 대해 염색된, TLE를 겪는 인간 환자로부터 절제된 기관형적 해마 뇌 슬라이스의 형광 이미지를 보여준다. PROX1 표지화는 치상회의 치상 과립 세포에서 관찰되었다. GFP 표지화는 또한, 치상 과립 세포에서 관찰되었다. 많은 세포가 PROX1 및 GFP의 공동표지화를 보여주었는데, 이것은 AAV9.GC.GFP가 치상 과립 세포를 견실하게 형질도입할 수 있다는 것을 암시하였다.
도 24는 인간 TLE 환자로부터 획득된 절제된 해마 조직에서 항-Grik2 miRNA 서열 (G9; 서열 번호: 68; n = 6명의 개체로부터 17개의 슬라이스) 또는 GI (서열 번호: 77; 2명의 개체로부터 2개의 슬라이스)를 인코딩하는 AAV9 발현 벡터를 이용한 GluK2 단백질 녹다운의 효능을 보여주는 산점도이다. GluK2 단백질 발현의 녹다운은 G9-인코딩 벡터로 처리된 인간 해마 조직의 5개 세트 중 5개에서 관찰되었다.
도 25는 TLE를 겪는 인간 환자로부터 절제되고 G9 (서열 번호: 68)를 인코딩하는 벡터로 처리된 기관형적 해마 슬라이스로부터 GluK2 단백질 발현을 보여주는 웨스턴 블롯 겔의 이미지를 보여준다. G9는 처리되지 않은 슬라이스에 비하여, GluK2 단백질 발현을 40% 감소시킬 수 있었다. GluK2 발현은 대조에 대해 정규화되었다.
도 26a-26c는 생리학적 상태 (ACSF) 하에 TLE를 겪는 인간 환자로부터 획득된 기관형적 해마 슬라이스로부터 예시적인 지역장 전위 기록 및 기록된 뇌전증모양 방전의 정량을 보여준다. 슬라이스는 항-Grik2 서열 G9-S1 (서열 번호: 775; 7개의 슬라이스; 도 26a)을 인코딩하는 벡터, 또는 스크램블 서열 GC 및 GFP 리포터 (서열 번호: 101; 6개의 슬라이스; 도 26b)를 인코딩하는 벡터로 처리되었다. 삽도는 더 높은 시간 해상도에서, 개별 뇌전증모양 방전의 전압 트레이스를 보여준다. 4명의 인간 TLE 환자로부터 획득된 슬라이스 전체에 대하여, G9-S1은 뇌전증모양 방전의 발생을 효과적으로 감소시키거나 또는 완전히 제거할 수 있었다 (도 26c; **p < 0.01).
도 27a-27d는 인간 TLE 환자로부터 절제된 기관형적 해마 슬라이스에서 Grik2 발현의 억제를 보여준다. DMTPV3 (서열 번호: 787) 또는 AAV.hSyn.GFP 대조 벡터로 처리된 인간 기관형적 해마 슬라이스에서 Grik2 유전자 발현의 산점도. DMTPV3은 qRT-PCR에 의해 계측될 때, Grik2 수준에서 실제적인 감소를 보여주었다 (도 27a). DMTPV3 또는 스크램블 대조 서열 GC (서열 번호: 101; 도 27b)를 인코딩하는 벡터로 치료를 받은 인간 TLE 환자로부터 획득된 절제된 기관형적 해마 뇌 슬라이스로부터 기록된 예시적인 전압 트레이스. 각 별표는 뇌전증모양 방전을 나타낸다. 삽도는 단일 뇌전증모양 방전의 확대된 트레이스를 보여준다. DMTPV3으로 처리된 슬라이스는 뇌전증모양 방전의 완전한 제거를 보여주었다 (도 27c). GC, G9, AAV9.hSyn.GFP, 또는 DMTPV3으로 처리된 슬라이스에서 기록된 뇌전증모양 방전의 주파수의 정량을 보여주는 막대 그래프 (도 27d). 첫 2개의 막대 (GC- 및 G9-처리 군에 상응)는 도 26c에서 도시된 것들과 동일하고 DMTPV3-처리 군과의 비교를 위해 포함된다는 점에 주의한다.
도 28은 대조 AAV9.hSyn.GFP 벡터 및 hSyn.G9-A-miR-30 벤치마크 벡터 (서열 번호: 775)에 비하여 발현 벡터 DMSPV1 (서열 번호: 811) 및 DMTPV8 (서열 번호: 813)로 처리된 생쥐 피질 뉴런에서 GluK2 발현의 백분율을 묘사하는 막대 그래프이다. 데이터는 평균 ± S.E.M으로서 제시된다. 발현 작제물 DMSPV1 및 DMPTV8은 생쥐 피질 뉴런에서 GluK2의 녹다운을, 벤치마크 hSyn.G9-A-miR-30 벡터와 비슷하게 야기하였다.
도 29는 대조 벡터 AAV9.hsyn.GFP (3.6E+9 MOI; n = 2), DMSPV1 (3.6E+9 또는 3.6E+8 MOI; n = 각 MOI에 대해 3), DMTPV8 (3.6E+9 또는 3.6E+8 MOI; n = 각 MOI에 대해 3)로 처리 전 (채워지지 않은 막대) 및 처리 후 (채워진 막대), 오픈 필드 상자에서 10 분의 자발적 탐색 동안 만성 뇌전증 생쥐에 의한 이동된 총 거리를 묘사하는 막대 그래프이다. DMSPV1 및 DMTPV8은 생쥐에서, 뇌전증발생에 대한 행동 프록시인 과운동 활성의 용량 의존성 감소를 발생시켰다.
도 30은 5'에서 3'로, 다음을 내포하는 본원 발명의 AAV 벡터의 계통도이다:
(a) AAV 5' ITR 서열 (예를 들면, 서열 번호: 746 및 747 중에서 하나);
(b) 프로모터 서열, 예컨대 예를 들면,
(i) hSyn 프로모터 (예를 들면, 서열 번호: 682, 683, 684 및 685 중에서 하나);
(ii) NeuN 프로모터 (예를 들면, 서열 번호: 686);
(iii) CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나);
(iv) NSE 프로모터 (예를 들면, 서열 번호: 692 또는 393);
(v) PDGF-베타 프로모터 (예를 들면, 서열 번호: 694-696 중에서 하나);
(vi) VGluT 프로모터 (예를 들면, 서열 번호: 697-701 중에서 하나);
(vii) SST 프로모터 (예를 들면, 서열 번호: 702 또는 703);
(viii) NPY 프로모터 (예를 들면, 서열 번호: 704);
(ix) VIP 프로모터 (예를 들면, 서열 번호: 705 또는 706);
(x) PV 프로모터 (예를 들면, 서열 번호: 707-709 중에서 하나);
(xi) GAD65 프로모터 (예를 들면, 서열 번호: 710-713 중에서 하나);
(xii) GAD67 프로모터 (예를 들면, 서열 번호: 714 또는 715);
(xiii) DRD1 프로모터 (예를 들면, 서열 번호: 716);
(xiv) DRD2 프로모터 (예를 들면, 서열 번호: 717 또는 718);
(xv) C1QL2 프로모터 (예를 들면, 서열 번호: 719);
(xvi) POMC 프로모터 (예를 들면, 서열 번호: 720);
(xvii) PROX1 프로모터 (예를 들면, 서열 번호: 721 또는 722);
(xviii) MAP1B 프로모터 (예를 들면, 서열 번호: 723-725 중에서 하나);
(xix) TUBA1A 프로모터 (예를 들면, 서열 번호: 726 또는 727);
(xx) U6 프로모터 (예를 들면, 서열 번호: 728-733 중에서 하나);
(xxi) H1 프로모터 (예를 들면, 서열 번호: 734);
(xxii) 7SK 프로모터 (예를 들면, 서열 번호: 735);
(xxiii) ApoE.hAAT 프로모터 (예를 들면, 서열 번호: 736);
(xxiv) CAG 프로모터 (예를 들면, 서열 번호: 737);
(xxv) CBA 프로모터 (예를 들면, 서열 번호: 738);
(xxvi) CK8 프로모터 (예를 들면, 서열 번호: 739);
(xxvii) MU1A 프로모터 (예를 들면, 서열 번호: 740);
(xxviii) EF1-알파 프로모터 (예를 들면, 서열 번호: 741); 및
(xxix) TBG 프로모터 (서열 번호: 742) 중에서 하나;
(c) 다음을 내포하는 스템 루프 서열:
(i) 5' 측부 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 및 768 중에서 하나);
(ii) 가이드 가닥 (예를 들면, 서열 번호: 1-100 중에서 하나) 또는 승객 가닥 서열 (예를 들면, 서열 번호: 1-100 중에서 하나에 실제적으로 또는 완전히 상보적인 서열)을 내포하는 5p 스템 루프 팔;
(iii) 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 및 770 중에서 하나); 및
(iv) 승객 가닥 (예를 들면, 서열 번호: 1-100 중에서 하나에 실제적으로 또는 완전히 상보적인 서열) 또는 가이드 가닥 서열 (예를 들면, 서열 번호: 1-100 중에서 하나)을 내포하는 3p 스템 루프 팔; 및
(v) 3' 측부 서열 (예를 들면, 서열 번호: 753, 754, 757, 760, 763, 766 및 769 중에서 하나);
(d) 3' 비번역 영역 (UTR; 예를 들면, 서열 번호: 750 및 751 중에서 하나); 그리고
(e) AAV 3' ITR 서열 (예를 들면, 서열 번호: 748 및 749 중에서 하나). 상기 요소 중에서 하나의 조합을 내포하는 AAV 벡터는 본원에서 개시된 방법에 따른 이용에 적합할 수 있다.
도 31은 3.6E+9 MOI의 대조 벡터 (CV; n = 3), 3.6E+9 MOI의 작제물 SMSPV4 (n = 4), 3.6E+8 MOI의 작제물 SMSPV4 (n = 4), 3.6E+9 MOI의 작제물 SMSPV5 (n = 4), 3.6E+8 MOI의 작제물 SMSPV5 (n = 4), 3.6E+9 MOI의 작제물 SMSPV6 (n = 4), 그리고 3.6E+8 MOI의 작제물 SMSPV6 (n = 4)으로 처리 전 (열린 막대) 및 처리 후 (채워진 막대), 오픈 필드 상자에서 10 분의 자발적 탐색 동안 만성 뇌전증 생쥐에 의한 이동된 총 거리를 보여주는 막대 그래프이다. SMSPV4, SMSPV5 및 SMSPV6으로 생쥐의 처리는 생쥐에서, 뇌전증 행동에 대한 프록시인 과운동을 감소시키지만, 대조 작제물은 그렇지 않았다. 프리 = 처리전; 포스트 = 처리후; E8 = 3.6E+8 MOI; E9 = 3.6E+9 MOI.
도 1a-1q는 세포에서 GluK2 단백질의 발현을 녹다운 (KD)시키기 위한 글루타민산염 이온성 수용체 카이네이트 유형 아단위 2 (Grik2) mRNA 안티센스 올리고뉴클레오티드 (ASO) 작제물의 확인과 사정을 보여준다. (도 1a) 중심 엔트로피 모형인 RNAfold에 의해 예측된 바와 같은 인간 Grik2 mRNA 변이체 1 (서열 번호: 115)의 예측된 이차 구조. (도 1b) Grik2 mRNA에 대한 복수의 항-Grik2 ASO의 결합의 예측된 영역. 결합의 예시적인 영역은 확인된 루프 영역 (1 = 루프 1 (서열 번호: 145); 2 = 루프 2 (서열 번호: 146); 3 = 루프 3 (서열 번호: 147); 4 = 루프 4 (서열 번호: 148); 5 = 루프 5 (서열 번호: 149); 6 = 루프 6 (서열 번호: 150); 7 = 비대합된 1 (서열 번호: 159); 8 = 비대합된 2 (서열 번호: 160); 9 = 루프 7 (서열 번호: 151); 10 = 루프 8 (서열 번호: 152); 11 = 루프 9 (서열 번호: 153); 12 = 루프 10 (서열 번호: 154); 13 = 비대합된 3 (서열 번호: 161); 14 = 루프 11 (서열 번호: 155); 15 = 비대합된 4 (서열 번호: 162); 16 = 비대합된 5 (서열 번호: 163); 17 = 루프 12 (서열 번호: 156); 18 = 루프 13 (서열 번호: 157); 및 19 = 루프 14 (서열 번호: 158)), 그리고 스템 유사 비대합 영역 (각각 서열 번호: 159-163에 상응하는 아라비아 숫자 15-19에 의해 지정됨)을 포함한다. (도 1c) Grik2 mRNA (서열 번호: 115)의 5' UTR 내에 결합의 예측된 영역에 맞춰 정렬된 항-Grik2 ASO 작용제 (서열 번호: 126; siRNA D3 (서열 번호: 48), siRNA XZ (서열 번호: 54), siRNA CY (서열 번호: 43), siRNA D1 (서열 번호: 46), siRNA GE (서열 번호: 65), siRNA CX (서열 번호: 42), siRNA Y0 (서열 번호: 55), siRNA TG (서열 번호: 23), siRNA D0 (서열 번호: 45), siRNA YB (서열 번호: 67), siRNA GF (서열 번호: 64), siRNA TD (서열 번호: 26), siRNA GH (서열 번호: 66), siRNA TE (서열 번호: 25), siRNA TJ (서열 번호: 21), siRNA TF (서열 번호: 24), siRNA YB/siSPOTR15 (서열 번호: 67), siRNA ZZ/siSPOTR16 (서열 번호: 100), siRNA GE/siSPOTR17 (서열 번호: 65), siRNA D3/siSPOTR18 (서열 번호: 48), siRNA CX/siSPOTR19 (서열 번호: 42), siRNA GF/siSPOTR20 (서열 번호: 64), siRNA GH/siSPOTR21 (서열 번호: 66), siRNA TJ/siSPOTR22 (서열 번호: 21), siRNA TG/siSPOTR23 (서열 번호: 23), siRNA TD/siSPOTR24 (서열 번호: 26) 및 siRNA TF/siSPOTR25 (서열 번호: 24)), 그리고 엑손 1의 코딩 서열 (CDS) (서열 번호: 129; siRNA CK (서열 번호: 29), siRNA TC (서열 번호: 28) 및 siRNA TC/siSPOTR1 (서열 번호: 28))의 계통도. (도 1d) Grik2 mRNA (서열 번호: 115)의 엑손 2 (서열 번호: 130) 내에 확인된 루프 1 영역 (서열 번호: 145)에 상대적으로 정렬된 예시적인 ASO 작용제 (G0; 서열 번호: 1)의 계통도. (도 1e) Grik2 mRNA (서열 번호: 115)의 엑손 10 (서열 번호: 138) 내에 확인된 루프 5 (서열 번호: 149) 및 루프 6 (서열 번호: 150) 영역에 상대적으로 정렬된 5개의 예시적인 ASO 서열 (GD (서열 번호: 7), MU (서열 번호: 96), MT (SEQ ID NO:99), MS (서열 번호: 99) 및 G3 (서열 번호: 8))의 계통도. (도 1f) Grik2 mRNA (서열 번호: 115)의 엑손 11 (서열 번호: 139)에 상대적으로 정렬된 예시적인 ASO 작용제 (MJ (서열 번호: 89), TH (서열 번호: 22), MI (서열 번호: 90), Y9 (서열 번호: 88), TK (서열 번호: 74), Y8 (서열 번호: 87), TI (서열 번호: 76), CU (서열 번호: 39) 및 Y7 (서열 번호: 62))를 보여주는 계통도. (도 1g) 이중-루시페라아제 리포터 검정에서 다양한 후보 ASO 작용제에 의한 Grik2 mRNA의 리포터 녹다운 퍼센트 (표 2를 또한 참조한다). (도 1h) 이중-루시페라아제 리포터 검정에서 다양한 후보 항-Grik2 ASO 작용제에 대한 비특이적 반딧불이 루시페라아제 (ffluc) 감소. (도 1i) 빈 대조 벡터 (관계 표적화된 비특이적 ffluc 녹다운)로부터 잔류 ffluc 발현의 함수로서 Grik2 mRNA 녹다운 퍼센트를 보여주는 산점도. (도 1j) 루시페라아제 리포터 검정에서 검사된 19 bp siRNA 모두에 대한 표적 개방 에너지 (kcal/mol)의 평균 (SEM 포함) 값의 막대 플롯. 데이터 막대 (중간 및 오른쪽 막대)는 리포터 유발 Gluk2 발현을 66% 초과하여 녹다운시킨 siRNA, 또는 발현을 66% 미만으로 녹다운시킨 siRNA에 의해 분리되었다. 10 kcal/mol 미만의 표적 개방 에너지를 갖는 siRNA의 강화된 클러스터는 66% 초과의 Gluk2 녹다운과 상관되었다. (도 1k) 루시페라아제 리포터 검정에서 검사된 바와 같은 19 bp 등가물의 녹다운 퍼센트에 대하여 21 bp 가이드 모두에 대한 표적 개방 에너지 (kcal/mol)의 평균 (SEM 포함) 값의 막대 플롯. 데이터 막대 (중간 및 오른쪽 막대)는 리포터 유발 Gluk2 발현을 66% 초과하여 녹다운시킨 등가 siRNA, 또는 발현을 66% 미만으로 녹다운시킨 등가 siRNA에 의해 분리되었다. 9.5 kcal/mol 미만의 표적 개방 에너지를 갖는 siRNA의 강화된 클러스터는 66% 초과 Gluk2 녹다운과 상관되었다. (도 1l) 루시페라아제 리포터 검정에서 검사된 19 bp siRNA 모두의 이중나선 형성의 에너지 (kcal/mol)의 평균 (SEM 포함)의 막대 플롯. 데이터 막대는 왼쪽에서 오른쪽으로: 검사된 19 bp siRNA 모두, 리포터 발현을 >66% 녹다운시킨 siRNA, 또는 리포터 발현을 <66% 녹다운시킨 siRNA를 나타낸다. (도 1m) 루시페라아제 리포터 검정에서 검사된 바와 같은 19 bp 등가물의 녹다운 퍼센트에 대하여 21 bp 가이드 모두에 대한 이중나선 형성의 에너지 (kcal/mol)의 평균 (SEM 포함) 값의 막대 플롯. 데이터 막대 (중간 및 오른쪽 막대)는 리포터 유발 Gluk2 발현을 66% 초과하여 녹다운시킨 등가 siRNA, 또는 발현을 66% 미만으로 녹다운시킨 등가 siRNA에 의해 분리되었다. (도 1n) 루시페라아제 리포터 검정에서 검사된 19 bp siRNA 모두에 대한 총 결합 에너지 (kcal/mol)의 평균 (SEM 포함)의 막대 플롯. 데이터 막대는 왼쪽에서 오른쪽으로: 검사된 19 bp siRNA 모두, 리포터 발현을 >66% 녹다운시킨 siRNA, 또는 리포터 발현을 <66% 녹다운시킨 siRNA를 나타낸다. (도 1o) 루시페라아제 리포터 검정에서 검사된 바와 같은 19 bp 등가물의 녹다운 퍼센트에 대하여 21 bp 가이드 모두에 대한 총 결합 에너지 (kcal/mol)의 평균 (SEM 포함) 값의 막대 플롯. 데이터 막대 (오른쪽, 중간)는 리포터 유발 Gluk2 발현을 66% 초과하여 녹다운시킨 등가 siRNA, 또는 발현을 66% 미만으로 녹다운시킨 등가 siRNA에 의해 분리되었다. (도 1p) 루시페라아제 리포터 검정에서 검사된 19 bp siRNA 각각에서 G 또는 C로서 확인된 염기의 퍼센트 (GC 함량)의 평균 (SEM 포함)의 막대 플롯. 데이터 막대는 왼쪽에서 오른쪽으로: 검사된 19 bp siRNA 모두, 리포터 발현을 >66% 녹다운시킨 siRNA, 또는 리포터 발현을 <66% 녹다운시킨 siRNA를 나타낸다. (도 1q) 루시페라아제 리포터 검정에서 검사된 바와 같은 19 bp 등가물의 녹다운 퍼센트에 대하여 21 bp 가이드 모두에 대한 GC 함량의 평균 (SEM 포함) 값의 막대 플롯. 데이터 막대 (오른쪽, 중간)는 리포터 유발 Gluk2 발현을 66% 초과하여 녹다운시킨 등가 siRNA, 또는 발현을 66% 미만으로 녹다운시킨 등가 siRNA에 의해 분리되었다.
도 2a-2j는 바이러스 벡터 매개 Grik2 mRNA 침묵에 의한 GluK2 녹다운의 검증을 보여준다. (도 2a-2d) 도 2a-2j에서 설명된 실험에서 활용된 예시적인 벡터. (도 2a) hSyn 프로모터 (서열 번호: 682)의 제어하에 대조 스크램블 서열 (서열 번호: 771)을 인코딩하는 렌티바이러스 벡터 (CM845)에 대한 예시적인 렌티바이러스 플라스미드 지도. (도 2b) U6 프로모터 (서열 번호: 772)의 제어하에 shRNA로서 Grik2 안티센스 서열 (G9; 서열 번호: 68)을 인코딩하는 렌티바이러스 벡터 (CM946)에 대한 예시적인 렌티바이러스 플라스미드 지도. (도 2c) hSyn 프로모터 (서열 번호: 683)의 제어하에 miRNA로서 Grik2 안티센스 서열 (G9; 서열 번호: 68)을 인코딩하는 렌티바이러스 벡터 (CM962)에 대한 예시적인 렌티바이러스 플라스미드 지도. (도 2d) hSyn 프로모터의 제어하에 GFP를 인코딩하는 AAV 벡터 (pAAV-hSyn-EGFP; 서열 번호: 682)에 대한 예시적인 플라스미드 지도. (도 2e) 렌티바이러스 (LV)-인간 시냅신 프로모터 (hSyn (서열 번호: 682))-녹색 형광 단백질 (GFP) 플라스미드 작제물로 감염된 배양된 쥐 해마 뉴런의 Brightfield (왼쪽 패널) 및 형광 (오른쪽 패널) 영상화. GFP 면역형광이 배양된 뉴런 중 >80%에서 관찰되었다. (도 2f) Grik2 안티센스 서열 (G9; 서열 번호: 68) 또는 대조 서열 (서열 번호: 771; hSyn 프로모터 (서열 번호: 682)의 제어하에)을 인코딩하는 짧은-헤어핀 RNA (LV-U6 (서열 번호: 772)-G9 (shRNA) 또는 마이크로RNA (LV-hSyn (서열 번호: 683)-G9 (miRNA) 작제물 중 어느 하나로 Grik2 mRNA의 렌티바이러스 매개 녹다운 이후에 쥐 해마 뉴런으로부터 획득된 GluK2 단백질 및 액틴의 웨스턴 블롯. (도 2g) 세포에서 AAV 매개 바이러스 형질도입에 이용된 AAV 발현 카세트의 계통도. (도 2h) 항-Grik2 ASO 서열 (G9; 서열 번호: 68) 또는 스크램블 대조 서열 (LV: 서열 번호: 771; AAV: GC - 서열 번호: 101)을 인코딩하는 렌티바이러스 또는 AAV9 벡터로 감염된 해마 뉴런에 대한 대조 조건에서 값에 대해 정규화된, GluK2 단백질 대 액틴의 상대적 수준을 나타내는 막대 그래프. (도 2i) 상이한 바이러스로 인코딩된 항-Grik2 ASO 서열 (G9 (서열 번호: 68); GI (서열 번호: 77); XY (서열 번호: 83); Y9 (서열 번호: 88); GG (서열 번호: 91)) 및 대조 서열 (GC; 서열 번호: 101)로 처리된 뮤린 일차 피질 뉴런에서, 웨스턴 블롯에 의해 검정될 때, GluK2 단백질의 상대적 수준을 보여주는 플롯. (도 2j) RT-qPCR에 의해 계측될 때, hSyn 프로모터 (서열 번호: 683)의 조절 제어하에 5개의 Grik2 mRNA 안티센스 올리고뉴클레오티드 (G9 (서열 번호: 68), GI (서열 번호: 77), Y9 (서열 번호: 88), XY (서열 번호: 83) 또는 MU (서열 번호: 96)) 중에서 하나, 또는 스크램블 대조 서열 (GC; 서열 번호: 101)을 인코딩하는 플라스미드 벡터와 함께 17,500개 세포/웰 (17.5k c/w)의 세포 밀도로 배양된 유도 만능 줄기 세포 (iPSC) 유래된 글루타민산성 뉴런 (GlutaNeurons)의 지질 기반 형질감염 5 일 후에 계측된 Grik2 mRNA 발현에서 배수 변화의 막대 플롯을 보여준다.
도 3a-3d는 뮤린 시험관내 모형에서 해마 뇌전증모양 활성에 대한 바이러스로 인코딩된 항-Grik2 ASO 작용제의 효과를 보여준다. (도 3a) 스크램블 서열 (서열 번호: 101)을 내포하는 AAV9-hSyn (서열 번호: 682)-GFP-스크램블 작제물로 감염된 기관형적 해마 뇌 슬라이스를 보여주는 형광 이미지. 상기 슬라이스는 해마의 치상회 (DG) 세포를 표지화하기 위해 프로스페로 호메오복스 단백질 1 (Prox1) 항체 (Millipore)로 면역염색되었다. (도 3b) 뮤린 기관형적 해마 슬라이스로부터 기록된 ED의 예시적인 세포외 전압 트레이스. (도 3c) hSyn 프로모터 (LV 및 AAV9- GC: 서열 번호: 682; AAV9-hSyn-G9: 서열 번호: 683), 또는 AAV9-GFP 대조 벡터의 제어하에 miRNA 작제물로서 항-Grik2 ASO 서열 (G9; 서열 번호: 68; ***, p< 0.001; **, p<0.01) 또는 스크램블 대조 서열 (AAV- GC (서열 번호: 101); LV-스크램블 (서열 번호: 771))을 인코딩하는 렌티바이러스 또는 AAV9 벡터로 감염된 뮤린 해마 슬라이스에서 ED의 주파수를 나타내는 막대 그래프. (도 3d) 표시된 AAV9-인코딩된 항-Grik2 ASO 작용제 (AAV9-hSyn (서열 번호: 683)-G9 (서열 번호: 68) - p = 0.0004; AAV9-hSyn (서열 번호: 683)-XY (서열 번호: 91) - p = 0.0008; AAV9-hSyn (서열 번호: 683)-GI (서열 번호: 85) - p = 0.0478); AAV9-hSyn (서열 번호: 683)-Y9 (서열 번호: 96); 및 AAV9-hSyn (서열 번호: 683)-GG (서열 번호: 91) 또는 대조 스크램블 서열 및 GFP 태그 (AAV9-hSyn (서열 번호: 682)-GFP-GC (서열 번호: 101))로 처리된 뮤린 기관형적 해마 슬라이스에서 ED의 주파수를 나타내는 막대 그래프.
도 4a-4f는 측두엽 뇌전증 (TLE)의 생체내 뮤린 모형에서 Grik2 표적화 ASO 작용제의 효능을 보여준다. (도 4a) 새로운 물체 인식 (NOR) 과제의 실험 설계에 대한 계통도. (도 4b) 바이러스로 인코딩된 스크램블 서열 (GC; 서열 번호: 101; AAV9-hSyn (서열 번호: 682)-GFP-GC) 또는 항-Grik2 서열 (G9; 서열 번호: 68; AAV9-hSyn (서열 번호: 683)-G9) 중 어느 하나로 주사 7 일 전에 또는 주사 15 일 후에 계측될 때, NOR 과제가 진행된 생쥐의 식별 지수 (DI)를 보여주는 막대 그래프. (도 4c) 바이러스로 인코딩된 스크램블 서열 (GC; 서열 번호: 101) 또는 항-Grik2 서열 (G9; 서열 번호: 68) 중 어느 하나로 주사 7 일 전에 또는 주사 15 일 후에 계측될 때, NOR 과제에서 생쥐에 의한 이동된 총 거리 (cm)를 보여주는 막대 그래프. (도 4d) 필로카르핀으로 처리 이후에 생쥐로부터 기록된 바와 같은, TLE의 필로카르핀 모형에서 유도된 전기사진술 발작의 예시적인 전압 트레이스. (도 4e) 바이러스로 인코딩된 스크램블 대조 서열 (GC; 서열 번호: 101; n = 3) 또는 바이러스로 인코딩된 항-Grik2 ASO 작용제 (G9; 서열 번호: 68; n = 4)로 처리된 생쥐에서 5 일에 걸쳐 누적 발작 지속 기간 (분)을 보여주는 막대 그래프. (도 4f) 바이러스로 인코딩된 스크램블 대조 서열 (GC; 서열 번호: 101; n = 5) 또는 바이러스로 인코딩된 항-Grik2 ASO 작용제 (G9; 서열 번호: 68; n = 6)로 처리된 생쥐에서 5 일에 걸쳐 발작의 누적 횟수를 보여주는 막대 그래프.
도 5는 AAV9 벡터에서 인코딩된 다양한 Grik2 mRNA 표적화 마이크로RNA 작제물의 녹다운 효능을 보여주는 막대 그래프이다. AAV9 벡터는 A-miR-30 (S1), E-miR-30 (S2), E-miR-155 (S3), E-miR-218 (S4) 및 E-miR-124 (S5)를 비롯한, 내인성 마이크로RNA로부터 5' 측부 영역, 마이크로RNA 루프 서열 및 3' 측부 영역을 내포하는 5개의 마이크로RNA 골격 중에서 하나를 통합한다. 검사된 안티센스 서열은 G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80), GU (서열 번호: 96), TO (서열 번호: 14), TK (서열 번호: 74), TH (서열 번호: 22), CQ (서열 번호: 35), XU (서열 번호: 51), XY (서열 번호: 83), Y9 (서열 번호: 88), YA (서열 번호: 63), GG (서열 번호: 91), G8 (서열 번호: 92), ME (서열 번호: 69), 그리고 MD (서열 번호: 70)이었다. 녹다운 효능은 "지질 단독" 대조에 비하여 Grik2 mRNA 중앙 배수 변화로서 표현된다. hSyn 프로모터 (서열 번호: 790)의 제어하에, miRNA 발현 플라스미드의 이러한 더 큰 패널은 유도 만능 줄기 세포 (iPSC) 유래된 글루타민산성 뉴런 (GlutaNeurons) 내로 형질감염되고, 그리고 RT-qPCR에 의해 Grik2 mRNA 수준을 감소시키는 능력에 대해 평가되었다. 비형질감염된 세포 (파선)와 비교하고, 그리고 중위 절대 편차 (MAD) = 2를 이용하여 기능적 작제물 (점선)을 확인할 때, 대다수의 작제물은 기능적인 것으로 결정되었다 (다시 말하면, 이들은 MAD 미만에서 녹다운 Grik2 mRNA를 나타낸다). 모든 검사된 작제물 중에서, GI (서열 번호: 77)-S2 (서열 번호: 798), MW (서열 번호: 80)-S4 (서열 번호: 799), MW-S5 (서열 번호: 800), 그리고 G9 (서열 번호: 68)-S5 (서열 번호: 801)가 Grik2 mRNA를 가장 높은 정도로 녹다운 (다시 말하면, 20% 이상 녹다운)시키는 것으로 밝혀졌다.
도 6a-6g는 내인성 miRNA로부터 5' 측부 영역, 마이크로RNA 루프 서열 및 3' 측부 영역을 내포하는 A-miR-30 (S1) 골격 내로 통합된 안티센스 가이드 서열을 내포하는 합성 AAV9-miRNA 작제물 입체형상의 계통도를 보여준다. 작제물 1 (도 6a)은 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 서열 (서열 번호: 790), miR-30 5' 측부 서열 (서열 번호: 752), G9의 항-Grik2 서열 (서열 번호: 68)에 실제적으로 상보적인 승객 가닥 서열, miR-30 루프 서열 (서열 번호: 758), G9의 가이드 서열 (서열 번호: 68), miR-30 3' 측부 서열 (서열 번호: 753), 토끼 베타-글로빈 (RBG) 폴리A 신호 (서열 번호: 792), 그리고 3' ITR 서열 (서열 번호: 789)을 내포하는 단일-miRNA, 단일 프로모터 작제물 (서열 번호: 775)이다. 작제물 2 (도 6b)는 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), C1ql2 프로모터 서열 (서열 번호: 791), hSyn 프로모터 서열 (서열 번호: 790), miR-30 5' 측부 서열 (서열 번호: 752), G9의 항-Grik2 서열 (서열 번호: 68)에 실제적으로 상보적인 승객 가닥 서열, miR-30 루프 서열 (서열 번호: 785), G9의 가이드 서열 (서열 번호: 68), miR-30 3' 측부 서열 (서열 번호: 753), RBG 폴리A 신호 (서열 번호: 792), 그리고 3' ITR 서열 (서열 번호: 748)을 내포하는 단일 miRNA, 이중 프로모터 작제물 (서열 번호: 777)이다. 작제물 3 (도 6c)은 hSyn 프로모터 (서열 번호: 790)에 인접한 5' 단부 (다시 말하면, 5')에서 야생형 AAV (wt)ITR 및 폴리A 서열의 하류에서 돌연변이체 ITR (mITR)을 내포하는 자가 상보성, 이중-miRNA (G9, 서열 번호: 68의 2개 사본), 단일 프로모터 작제물 (서열 번호: 779)이다. 작제물 4 (도 6d; 서열 번호: 781)는 hSyn 프로모터 (서열 번호: 790)가 mITR에 인접하고 폴리A 서열이 wtITR에 인접하다는 점을 제외하고, 작제물 3과 유사하다. 작제물 5 (서열 번호: 783) 및 작제물 6 (서열 번호: 784)은 이들 작제물이 동일한 miRNA 서열 (예를 들면, G9, 서열 번호: 68; 작제물 5)의 3개 사본 또는 상이한 miRNA 서열 (도 6e; 작제물 6; G9, GI (서열 번호: 77), MU (서열 번호: 96))의 3개 사본을 내포하도록, 프리-miR 스템 루프 구조 (5' 측부, 스템 루프 및 3' 측부)가 3회 연쇄체화된다는 점을 제외하고, 작제물 1과 유사하다. 작제물 7 (도 6f; 서열 번호: 804)은 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 서열 (서열 번호: 790), miR-30 5' 측부 서열 (서열 번호: 752), G9의 항-Grik2 서열 (서열 번호: 68)에 실제적으로 상보적인 승객 가닥 서열, miR-30 루프 서열 (서열 번호: 758), G9의 가이드 서열 (서열 번호: 68), miR-30 3' 측부 서열 (서열 번호: 753), RBG 폴리A 신호 (서열 번호: 792), 비코딩 스터퍼 서열, 그리고 3' ITR 서열 (서열 번호: 789)을 내포하는 단일-miRNA, 단일 프로모터 작제물이다. 작제물 8 (도 6g; 서열 번호: 810)은 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 서열 (서열 번호: 790), E-miR-124-3 5' 측부 서열 (서열 번호: 768), G9의 안티센스 서열 (서열 번호: 68)에 상보적인 센스 승객 가닥 서열, E-miR-124-3 루프 서열 (서열 번호: 770), G9의 안티센스 가이드 서열, E-miR-124-3 3' 측부 서열 (서열 번호: 769), RBG 폴리A 신호 (서열 번호: 792), 비코딩 스터퍼 서열, 그리고 3' ITR 서열 (서열 번호: 789)을 내포하는 단일-miRNA, 단일 프로모터 작제물이다.
도 7은 도 6a-6e에서 설명된 단일- 및 이중-miRNA 발현 작제물 (작제물 1-6)의 알칼리성 아가로즈 겔 전기이동 분석을 보여주는 사진이다. 단일 프로모터 및 단일 miRNA 카세트 (예상된 길이: 1.5 kb)를 인코딩하는 플라스미드로부터 생성된 벡터의 유전체 내용물은 단독적으로 (1.5 kb), 이중으로 (3.0 kb) 및 삼중으로 (4.5 kb) 포장된 유전체의 혼합물로 구성되는 것으로 밝혀졌다. 레인 번호는 아래의 벡터 작제물에 상응한다: 1 = 작제물 1 (서열 번호: 775); 2 = 작제물 2 (서열 번호: 777); 3 = 작제물 3 (서열 번호: 779); 4 = 작제물 4 (서열 번호: 781); 5 = 작제물 5 (서열 번호: 783); 6 = 작제물 6 (서열 번호: 784).
도 8a-8g는 AAV 벡터용으로 적합한 이중 프로모터를 갖는 AAV9 이중-miRNA 발현 작제물의 계통도를 보여준다. 도 8a는 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 (서열 번호: 790), E-miR-124-3 5' 측부 서열 (서열 번호: 768), G9의 안티센스 서열 (서열 번호: 68)에 상보적인 센스 승객 ("P") 가닥 서열, E-miR-124-3 루프 서열 (서열 번호: 770), G9의 안티센스 가이드 ("G") 서열, E-miR-124-3 3' 측부 서열 (서열 번호: 769), BGH 폴리A 서열 (서열 번호: 793), CaMKII 프로모터 서열 (서열 번호: 802), E-miR-30 5' 측부 서열 (서열 번호: 759), GI (서열 번호: 77)에 상보적인 센스 승객 가닥 서열, E-miR-30 루프 서열 (서열 번호: 761), GI의 안티센스 가이드 서열 (서열 번호: 77), E-miR-30 3' 측부 서열 (서열 번호: 760), RBG 폴리A 서열 (서열 번호: 792), 그리고 3' ITR 서열 (서열 번호: 748)을 내포하는 이중-miRNA 이중 프로모터 벡터 (DMTPV1) 발현 작제물 (서열 번호: 785)을 보여준다. 도 8b는 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 (서열 번호: 790), E-miR-124-3 5' 측부 서열 (서열 번호: 768), G9의 안티센스 서열 (서열 번호: 68)에 상보적인 센스 승객 가닥 서열, E-miR-124-3 루프 서열 (서열 번호: 770), G9의 안티센스 가이드 서열, E-miR-124-3 3' 측부 서열 (서열 번호: 769), BGH 폴리A 서열 (서열 번호: 793), CaMKII 프로모터 서열 (서열 번호: 802), E-miR-218 5' 측부 서열 (서열 번호: 765), MW (서열 번호: 80)에 상보적인 센스 승객 가닥 서열, E-miR-218 루프 서열 (서열 번호: 767), MW의 안티센스 가이드 서열 (서열 번호: 80), E-miR-218 3' 측부 서열 (서열 번호: 766), RBG 폴리A 서열 (서열 번호: 792), 그리고 3' ITR 서열 (서열 번호: 748)을 내포하는 이중 siRNA 발현 작제물 (DMTPV2, 서열 번호: 786)을 보여준다. 도 8c는 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 (서열 번호: 790), E-miR-30 5' 측부 서열 (서열 번호: 759), GI (서열 번호: 77)에 상보적인 센스 승객 가닥 서열, E-miR-30 루프 서열 (서열 번호: 761), GI의 안티센스 가이드 서열 (서열 번호: 77), E-miR-30 3' 측부 서열 (서열 번호: 760), BGH 폴리A 서열 (서열 번호: 793), CaMKII 프로모터 서열 (서열 번호: 802), E-miR-124-3 5' 측부 서열 (서열 번호: 768), G9의 안티센스 서열 (서열 번호: 68)에 상보적인 센스 승객 가닥 서열, E-miR-124-3 루프 서열 (서열 번호: 770), G9의 안티센스 가이드 서열, E-miR-124-3 3' 측부 서열 (서열 번호: 769), RBG 폴리A 서열 (서열 번호: 792), 그리고 3' ITR 서열 (서열 번호: 748)을 내포하는 이중 siRNA 발현 작제물 (DMTPV3, 서열 번호: 787)을 보여준다. 도 8d는 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 (서열 번호: 790), E-miR-30 5' 측부 서열 (서열 번호: 759), GI (서열 번호: 77)에 상보적인 센스 승객 가닥 서열, E-miR-30 루프 서열 (서열 번호: 761), GI의 안티센스 가이드 서열 (서열 번호: 77), E-miR-30 3' 측부 서열 (서열 번호: 760), BGH 폴리A 서열 (서열 번호: 793), CaMKII 프로모터 서열 (서열 번호: 802), E-miR-124-3 5' 측부 서열 (서열 번호: 768), MW의 안티센스 서열 (서열 번호: 80)에 상보적인 센스 승객 가닥 서열, E-miR-124-3 루프 서열 (서열 번호: 770), MW의 안티센스 가이드 서열 (서열 번호: 80), E-miR-124-3 3' 측부 서열 (서열 번호: 769), RBG 폴리A 서열 (서열 번호: 792), 그리고 3' ITR 서열 (서열 번호: 748)을 내포하는 이중 siRNA 발현 작제물 (DMTPV4, 서열 번호: 788)을 보여준다. 도 8e는 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 (서열 번호: 790), E-miR-30 5' 측부 서열 (서열 번호: 759), GI (서열 번호: 77)에 상보적인 센스 승객 가닥 서열, E-miR-30 루프 서열 (서열 번호: 761), GI의 안티센스 가이드 서열 (서열 번호: 77), E-miR-30 3' 측부 서열 (서열 번호: 760), BGH 폴리A 서열 (서열 번호: 793), CaMKII 프로모터 서열 (서열 번호: 802), E-miR-218 5' 측부 서열 (서열 번호: 765), MW의 안티센스 서열 (서열 번호: 80)에 상보적인 센스 승객 가닥 서열, E-miR-218 루프 서열 (서열 번호: 767), MW의 안티센스 가이드 서열 (서열 번호: 80), E-miR-218 3' 측부 서열 (서열 번호: 766), RBG 폴리A 서열 (서열 번호: 792), 그리고 3' ITR 서열 (서열 번호: 748)을 내포하는 이중 siRNA 발현 작제물 (DMTPV5)을 보여준다. 도 8f는 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 (서열 번호: 790), E-miR-30 5' 측부 서열 (서열 번호: 759), GI (서열 번호: 77)에 상보적인 센스 승객 가닥 서열, E-miR-30 루프 서열 (서열 번호: 761), GI의 안티센스 가이드 서열 (서열 번호: 77), E-miR-30 3' 측부 서열 (서열 번호: 760), E-miR-218 5' 측부 서열 (서열 번호: 765), MW의 안티센스 서열 (서열 번호: 80)에 상보적인 센스 승객 가닥 서열, E-miR-218 루프 서열 (서열 번호: 767), MW의 안티센스 가이드 서열 (서열 번호: 80), E-miR-218 3' 측부 서열 (서열 번호: 766), RBG 폴리A 서열 (서열 번호: 792), 비코딩 스터퍼 서열, 그리고 3' ITR 서열 (서열 번호: 748)을 내포하는 이중 siRNA 발현 작제물 (DMTPV6)을 보여준다. 도 8g는 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 (서열 번호: 790), E-miR-30 5' 측부 서열 (서열 번호: 759), GI (서열 번호: 77)에 상보적인 센스 승객 가닥 서열, E-miR-30 루프 서열 (서열 번호: 761), GI의 안티센스 가이드 서열 (서열 번호: 77), E-miR-30 3' 측부 서열 (서열 번호: 760), BGH 폴리A 서열 (서열 번호: 793), CaMKII 프로모터 서열 (서열 번호: 802), E-miR-124-3 5' 측부 서열 (서열 번호: 768), G9의 안티센스 가이드 서열, E-miR-124-3 루프 서열 (서열 번호: 770), G9의 안티센스 서열 (서열 번호: 68)에 상보적인 센스 승객 가닥 서열, E-miR-124-3 3' 측부 서열 (서열 번호: 769), RBG 폴리A 서열 (서열 번호: 792), 그리고 3' ITR 서열 (서열 번호: 748)을 내포하는 이중 siRNA 발현 작제물 (DMTPV7)을 보여준다.
도 9a 및 9b는 단일-siRNA 벡터 작제물 (도 9a; G9, 서열 번호: 68 - 작제물 1 (서열 번호: 775); GC, 서열 번호: 101) 및 이중-miRNA 벡터 작제물 (도 9b; DMTPV1-4; 각각, 서열 번호: 785-788)로부터 생산된 벡터의 cDNA의 알칼리성 아가로오스 겔 분석을 보여주는 사진이다. 4개의 이중-miRNA 벡터 작제물 전체에 대하여 단일 띠는 이중 발현 작제물의 벡터가 AAV9 벡터에서 단독적으로 포장된다는 것을 암시한다.
도 10은 qPCR에 의해 계측될 때, 단독적으로 또는 상이한 miRNA 서열을 내포하는 다른 단일-miRNA AAV9 작제물 (G9-S1 (서열 번호: 775), GI-S1 (서열 번호: 796), GI-S2 (서열 번호: 798), MW-S4 (서열 번호: 799), G9-S5 (서열 번호: 800) 또는 이들의 조합)과 조합으로 전달된 단일-miRNA AAV9 작제물을 이용한 GluK2 단백질 녹다운의 시험관내 효능을 나타내는 막대 그래프를 보여준다. 두 가지 상이한 항-Grik2 miRNA 서열 (둘 모두 hSyn 프로모터 (서열 번호: 790)의 제어하에)의 조합으로 형질감염된 GlutaNeurons가 단일 유형의 항-Grik2 miRNA 서열로 형질감염된 GlutaNeurons와 유사한 GluK2 단백질 녹다운을 보여주었는데, 이것은 GluK2 발현을 녹다운시키기 위한, Grik2에 대한 하나 초과의 고유한 안티센스 가이드 서열을 인코딩하는 벡터의 이용을 뒷받침하였다. 녹다운 효능은 "지질 단독" 대조군에 비하여 중앙 Grik2 mRNA 수준 배수 변화에서 배수 변화로서 계측되었다.
도 11은 hSyn 프로모터 (서열 번호: 790)의 제어하에 상이한 단일-miRNA AAV9 발현 벡터 (GC (서열 번호: 101); G9-S1 (서열 번호: 775), GI-S1 (서열 번호: 796), 또는 G9-S1 + GI-S1)의 조합으로 형질감염된 무억제성 뮤린 기관형적 해마 슬라이스에서 뇌전증모양 활성의 주파수의 막대 그래프를 보여준다. miRNA 작제물, G9-S1 및 GI-S1의 조합은 개별적으로 각 벡터와 동등한 정도의 뇌전증모양 활성 억제를 보여주었는데, 이것은 해마 회로에서 뇌전증모양 활성을 억제하기 위한, Grik2에 대한 하나 초과의 고유한 안티센스 가이드 서열의 이용을 뒷받침하였다.
도 12는 hSyn.GI (서열 번호: 77).S2 (서열 번호: 798), hSyn.MW (서열 번호: 80).S4 (서열 번호: 799), hSyn.MW.S5 (서열 번호: 800), hSyn.G9 (서열 번호: 68).S5 (서열 번호: 801), CaMKII.GI.S4, CaMKII.MW.S5, CaMKII.G9.S5, DMTPV1 (서열 번호: 785), DMTPV2 (서열 번호: 786), DMTPV3 (서열 번호: 787) 및 DMTPV4 (서열 번호: 788)를 비롯한, 여러 안티센스 작제물 중에서 하나를 이용하여 GlutaNeurons에서 Grik2의 AAV9 벡터 매개 녹아웃 이후에, qPCR에 의해 계측될 때, Grik2 mRNA의 수준을 나타내는 막대 그래프를 보여준다. mRNA 수준은 RNA를 생산하지 않는 전장 유전체를 내포하는 AAV.null 벡터로 형질도입된 GlutaNeurons와 비교되었다.
도 13a 및 13b는 필로카르핀 및 여러 단일-miRNA 벡터 작제물 (GC (서열 번호: 101), G9-S1 (서열 번호: 775), 또는 GI-S1 (서열 번호: 796)) 중에서 하나로 처리된 생쥐를 이용하여 수행된 오픈 필드 시험의 결과를 보여준다. 도 13a는 오픈 필드에서 단일 생쥐에 대한 추적된 운동의 예시적인 트레이스를 보여준다. 도 13b는 항-Grik2 miRNA 서열을 인코딩하는 AAV9 벡터로 처리된 생쥐에 의한 오픈 필드 시험에서 이동된 총 거리를 나타내는 막대 그래프를 보여준다. 비-뇌전증 생쥐 (다시 말하면, 필로카르핀으로 처리되지 않은 생쥐; n=20) 및 GC, G9-S1 또는 GI-S1 (각각, n=9, n=8, n=9)으로 처리된 만성 뇌전증 생쥐. 주사전 및 주사후 데이터는 만 휘트니 검정, *p<0.05 및 **p<0.01을 이용하여 비교되었다. G9 및 GI에서 과운동의 유의미한 감소에 주목한다.
도 14는 항-Grik2 작제물 G9-S1 (서열 번호: 775), GI-S1 (서열 번호: 796), 또는 스크램블 대조 작제물 GC (서열 번호: 101)를 인코딩하는 AAV9 벡터로 처리된 필로카르핀-처리 생쥐 (각각, n=5, n=5, n=5)에서 하루에 뇌전증 발작의 총수를 보여주는 막대 그래프이다. G9-S1 및 GI-S1 미가공 데이터는 일원 변량 분석 검정, p<0.01을 이용하여 GC와 비교되었다. G9-S1 및 GI-S1에서 발작의 억제에 주목한다.
도 15는 상이한 용량에서 항-Grik2 작제물 G9 (서열 번호: 68)를 인코딩하는 AAV9 벡터로 처리된 생쥐에 의한 오픈 필드 시험에서 이동된 총 거리를 보여주는 막대 그래프이다. 상이한 용량의 G9: G9/1, G9/10, G9/100 및 G9/1000으로 처리된 만성 뇌전증 생쥐 (각각, n=8, n=5, n=5, n=5). GC는 대조 작제물 (n=9)이다. 주사전 및 주사후 데이터는 만 휘트니 검정, *p<0.05 및 **p<0.01을 이용하여 비교되었다. G9 및 G9/10의 유사한 효과에 주목한다.
도 16은 여러 용량: G9/1, G9/10, G9/100 및 G9/1000 중 하나에서 항-Grik2 작제물 G9-S1/G9 (서열 번호: 775)를 인코딩하는 AAV9 벡터로 처리된 필로카르핀-처리 생쥐 (각각, n=6, n=4, n=2, n=2)에서 하루에 뇌전증 발작의 총수를 보여주는 막대 그래프이다. G9 및 G9/10에서는 유사한 효과가 달성되지만, G9/1000에서는 그렇지 않다는 점에 주목한다.
도 17은 여러 이중-miRNA, 이중 프로모터 작제물 (DMTPV1-4) 중에서 하나를 인코딩하는 AAV9 벡터로 처리된 필로카르핀-처리 생쥐에 의한 오픈 필드 시험에서 이동된 총 거리를 보여주는 막대 그래프이다. 비-뇌전증 생쥐 (n=20) 및 DMTPV1 (서열 번호: 785), DMTPV2 (서열 번호: 786), DMTPV3 (서열 번호: 787) 또는 DMTPV4 (서열 번호: 788)로 처리된 만성 뇌전증 생쥐 (각각, n=6, n=5, n=6 및 n=6). 주사전 및 주사후 데이터는 만 휘트니 검정, *p<0.05 및 **p<0.01을 이용하여 비교되었다. DMTPV3 및 DMTPV4에서 과운동의 유의미한 감소에 주목한다.
도 18은 필로카르핀-처리 생쥐에서 하루에 자연발생적 뇌전증 발작의 횟수와 대비하여 오픈 필드 시험에서 이동된 총 거리 (cm)를 보여주는 산점도이다. 회귀 분석은 과운동 및 발작 감수성 사이의 유의미한 상관을 증명한다 (R2 = 0.7388, p < 0.0001).
도 19는 G9 (서열 번호: 68), GI (서열 번호: 77), DMTPV1 (서열 번호: 785), DMTPV2 (서열 번호: 786), DMTPV3 (서열 번호: 787) 및 DMTPV4 (서열 번호: 788)를 비롯한 여러 항-Grik2 miRNA 서열 중에서 하나, 그리고 대조 AAV9.hSyn.GFP 벡터로 GlutaNeurons의 형질도입 이후에 Grik2 mRNA 발현을 보여주는 막대 그래프이다. 모든 검사된 이중-miRNA 작제물은 RNA 염기서열분석을 이용하여 계측될 때, GlutaNeurons에서 Grik2 mRNA 수준을 감소시켰다. 배수 변화는 대조에 상대적이다.
도 20은 단일 항-Grik2 miRNA 서열 G9 (서열 번호: 68)를 인코딩하는 벡터, 이중-miRNA 벡터 DMTPV3 (서열 번호: 787)으로 처리된 생쥐, 또는 대조 AAV9.hSyn.GFP 벡터로 처리된 생쥐의 오픈 필드 시험에서 운동을 보여주는 막대 그래프이다. WT 생쥐는 별개의 대조로서 이용되었다. 프리 = 처리전; 포스트 = 처리후. G9- 및 DMTPV3-인코딩 벡터 둘 모두 생쥐에서 과운동 활성을 유의미하게 억제하였는데 (p < 0.01; 만 휘트니 검정), 이것은 과운동의 억제에 대한 이들 벡터의 견실한 효과를 암시하였다.
도 21은 DMTPV3 (3.6x1010개 GC/뇌), DMTPV3/10 (3.6x109개 GC/뇌), DMTPV3/100 (3.6x108개 GC/뇌) 및 DMTPV3/1000 (3.6x107개 GC/뇌)을 비롯한, 변하는 용량의 DMTPV3 (서열 번호: 787)으로 처리된 생쥐의 오픈 필드 시험에서 과운동 활성의 용량 의존성 감소를 보여주는 막대 그래프이다. 대조 생쥐는 AAV9.hSyn.GFP 벡터 (3.6x1010개 GC/뇌)로 처리되었다. 프리 = 처리전; 포스트 = 처리후. DMTPV3 및 DMTPV3/10 투약으로 처리된 생쥐는 대조 생쥐에 비하여 과운동 활성에서 유의미한 감소를 보여주었다 (p < 0.01; 만 휘트니 검정).
도 22는 항-Grik2 단일-miRNA 작제물 G9 (서열 번호: 68) 또는 GI (서열 번호: 77), 또는 이중-miRNA 작제물 DMTPV3 (서열 번호: 787)을 인코딩하는 벡터로 더욱 처리된 필로카르핀-처리 생쥐에서 하루에 발작의 횟수를 보여주는 막대 그래프이다. 생쥐는 또한, 스크램블 RNA 서열 GC (서열 번호: 101) 또는 AAV9.hSyn.GFP를 인코딩하는 대조 벡터로 처리되었다. G9, GI 및 DMTPV3으로 처리된 생쥐는 하루에 발작의 횟수에서 유의미한 감소를 보여주었는데, DMTPV가 G9 및 GI와 비교하여 더 큰 감소를 보여주었다 (* p < 0.05; ** p < 0.01; 만 휘트니 검정).
도 23은 DIV 1 이후에 AAV9.GC(SEQ 식별 번호: 101).GFP로 감염되고 치상 과립 세포의 마커 (PROX1)에 대해 염색된, TLE를 겪는 인간 환자로부터 절제된 기관형적 해마 뇌 슬라이스의 형광 이미지를 보여준다. PROX1 표지화는 치상회의 치상 과립 세포에서 관찰되었다. GFP 표지화는 또한, 치상 과립 세포에서 관찰되었다. 많은 세포가 PROX1 및 GFP의 공동표지화를 보여주었는데, 이것은 AAV9.GC.GFP가 치상 과립 세포를 견실하게 형질도입할 수 있다는 것을 암시하였다.
도 24는 인간 TLE 환자로부터 획득된 절제된 해마 조직에서 항-Grik2 miRNA 서열 (G9; 서열 번호: 68; n = 6명의 개체로부터 17개의 슬라이스) 또는 GI (서열 번호: 77; 2명의 개체로부터 2개의 슬라이스)를 인코딩하는 AAV9 발현 벡터를 이용한 GluK2 단백질 녹다운의 효능을 보여주는 산점도이다. GluK2 단백질 발현의 녹다운은 G9-인코딩 벡터로 처리된 인간 해마 조직의 5개 세트 중 5개에서 관찰되었다.
도 25는 TLE를 겪는 인간 환자로부터 절제되고 G9 (서열 번호: 68)를 인코딩하는 벡터로 처리된 기관형적 해마 슬라이스로부터 GluK2 단백질 발현을 보여주는 웨스턴 블롯 겔의 이미지를 보여준다. G9는 처리되지 않은 슬라이스에 비하여, GluK2 단백질 발현을 40% 감소시킬 수 있었다. GluK2 발현은 대조에 대해 정규화되었다.
도 26a-26c는 생리학적 상태 (ACSF) 하에 TLE를 겪는 인간 환자로부터 획득된 기관형적 해마 슬라이스로부터 예시적인 지역장 전위 기록 및 기록된 뇌전증모양 방전의 정량을 보여준다. 슬라이스는 항-Grik2 서열 G9-S1 (서열 번호: 775; 7개의 슬라이스; 도 26a)을 인코딩하는 벡터, 또는 스크램블 서열 GC 및 GFP 리포터 (서열 번호: 101; 6개의 슬라이스; 도 26b)를 인코딩하는 벡터로 처리되었다. 삽도는 더 높은 시간 해상도에서, 개별 뇌전증모양 방전의 전압 트레이스를 보여준다. 4명의 인간 TLE 환자로부터 획득된 슬라이스 전체에 대하여, G9-S1은 뇌전증모양 방전의 발생을 효과적으로 감소시키거나 또는 완전히 제거할 수 있었다 (도 26c; **p < 0.01).
도 27a-27d는 인간 TLE 환자로부터 절제된 기관형적 해마 슬라이스에서 Grik2 발현의 억제를 보여준다. DMTPV3 (서열 번호: 787) 또는 AAV.hSyn.GFP 대조 벡터로 처리된 인간 기관형적 해마 슬라이스에서 Grik2 유전자 발현의 산점도. DMTPV3은 qRT-PCR에 의해 계측될 때, Grik2 수준에서 실제적인 감소를 보여주었다 (도 27a). DMTPV3 또는 스크램블 대조 서열 GC (서열 번호: 101; 도 27b)를 인코딩하는 벡터로 치료를 받은 인간 TLE 환자로부터 획득된 절제된 기관형적 해마 뇌 슬라이스로부터 기록된 예시적인 전압 트레이스. 각 별표는 뇌전증모양 방전을 나타낸다. 삽도는 단일 뇌전증모양 방전의 확대된 트레이스를 보여준다. DMTPV3으로 처리된 슬라이스는 뇌전증모양 방전의 완전한 제거를 보여주었다 (도 27c). GC, G9, AAV9.hSyn.GFP, 또는 DMTPV3으로 처리된 슬라이스에서 기록된 뇌전증모양 방전의 주파수의 정량을 보여주는 막대 그래프 (도 27d). 첫 2개의 막대 (GC- 및 G9-처리 군에 상응)는 도 26c에서 도시된 것들과 동일하고 DMTPV3-처리 군과의 비교를 위해 포함된다는 점에 주의한다.
도 28은 대조 AAV9.hSyn.GFP 벡터 및 hSyn.G9-A-miR-30 벤치마크 벡터 (서열 번호: 775)에 비하여 발현 벡터 DMSPV1 (서열 번호: 811) 및 DMTPV8 (서열 번호: 813)로 처리된 생쥐 피질 뉴런에서 GluK2 발현의 백분율을 묘사하는 막대 그래프이다. 데이터는 평균 ± S.E.M으로서 제시된다. 발현 작제물 DMSPV1 및 DMPTV8은 생쥐 피질 뉴런에서 GluK2의 녹다운을, 벤치마크 hSyn.G9-A-miR-30 벡터와 비슷하게 야기하였다.
도 29는 대조 벡터 AAV9.hsyn.GFP (3.6E+9 MOI; n = 2), DMSPV1 (3.6E+9 또는 3.6E+8 MOI; n = 각 MOI에 대해 3), DMTPV8 (3.6E+9 또는 3.6E+8 MOI; n = 각 MOI에 대해 3)로 처리 전 (채워지지 않은 막대) 및 처리 후 (채워진 막대), 오픈 필드 상자에서 10 분의 자발적 탐색 동안 만성 뇌전증 생쥐에 의한 이동된 총 거리를 묘사하는 막대 그래프이다. DMSPV1 및 DMTPV8은 생쥐에서, 뇌전증발생에 대한 행동 프록시인 과운동 활성의 용량 의존성 감소를 발생시켰다.
도 30은 5'에서 3'로, 다음을 내포하는 본원 발명의 AAV 벡터의 계통도이다:
(a) AAV 5' ITR 서열 (예를 들면, 서열 번호: 746 및 747 중에서 하나);
(b) 프로모터 서열, 예컨대 예를 들면,
(i) hSyn 프로모터 (예를 들면, 서열 번호: 682, 683, 684 및 685 중에서 하나);
(ii) NeuN 프로모터 (예를 들면, 서열 번호: 686);
(iii) CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나);
(iv) NSE 프로모터 (예를 들면, 서열 번호: 692 또는 393);
(v) PDGF-베타 프로모터 (예를 들면, 서열 번호: 694-696 중에서 하나);
(vi) VGluT 프로모터 (예를 들면, 서열 번호: 697-701 중에서 하나);
(vii) SST 프로모터 (예를 들면, 서열 번호: 702 또는 703);
(viii) NPY 프로모터 (예를 들면, 서열 번호: 704);
(ix) VIP 프로모터 (예를 들면, 서열 번호: 705 또는 706);
(x) PV 프로모터 (예를 들면, 서열 번호: 707-709 중에서 하나);
(xi) GAD65 프로모터 (예를 들면, 서열 번호: 710-713 중에서 하나);
(xii) GAD67 프로모터 (예를 들면, 서열 번호: 714 또는 715);
(xiii) DRD1 프로모터 (예를 들면, 서열 번호: 716);
(xiv) DRD2 프로모터 (예를 들면, 서열 번호: 717 또는 718);
(xv) C1QL2 프로모터 (예를 들면, 서열 번호: 719);
(xvi) POMC 프로모터 (예를 들면, 서열 번호: 720);
(xvii) PROX1 프로모터 (예를 들면, 서열 번호: 721 또는 722);
(xviii) MAP1B 프로모터 (예를 들면, 서열 번호: 723-725 중에서 하나);
(xix) TUBA1A 프로모터 (예를 들면, 서열 번호: 726 또는 727);
(xx) U6 프로모터 (예를 들면, 서열 번호: 728-733 중에서 하나);
(xxi) H1 프로모터 (예를 들면, 서열 번호: 734);
(xxii) 7SK 프로모터 (예를 들면, 서열 번호: 735);
(xxiii) ApoE.hAAT 프로모터 (예를 들면, 서열 번호: 736);
(xxiv) CAG 프로모터 (예를 들면, 서열 번호: 737);
(xxv) CBA 프로모터 (예를 들면, 서열 번호: 738);
(xxvi) CK8 프로모터 (예를 들면, 서열 번호: 739);
(xxvii) MU1A 프로모터 (예를 들면, 서열 번호: 740);
(xxviii) EF1-알파 프로모터 (예를 들면, 서열 번호: 741); 및
(xxix) TBG 프로모터 (서열 번호: 742) 중에서 하나;
(c) 다음을 내포하는 스템 루프 서열:
(i) 5' 측부 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 및 768 중에서 하나);
(ii) 가이드 가닥 (예를 들면, 서열 번호: 1-100 중에서 하나) 또는 승객 가닥 서열 (예를 들면, 서열 번호: 1-100 중에서 하나에 실제적으로 또는 완전히 상보적인 서열)을 내포하는 5p 스템 루프 팔;
(iii) 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 및 770 중에서 하나); 및
(iv) 승객 가닥 (예를 들면, 서열 번호: 1-100 중에서 하나에 실제적으로 또는 완전히 상보적인 서열) 또는 가이드 가닥 서열 (예를 들면, 서열 번호: 1-100 중에서 하나)을 내포하는 3p 스템 루프 팔; 및
(v) 3' 측부 서열 (예를 들면, 서열 번호: 753, 754, 757, 760, 763, 766 및 769 중에서 하나);
(d) 3' 비번역 영역 (UTR; 예를 들면, 서열 번호: 750 및 751 중에서 하나); 그리고
(e) AAV 3' ITR 서열 (예를 들면, 서열 번호: 748 및 749 중에서 하나). 상기 요소 중에서 하나의 조합을 내포하는 AAV 벡터는 본원에서 개시된 방법에 따른 이용에 적합할 수 있다.
도 31은 3.6E+9 MOI의 대조 벡터 (CV; n = 3), 3.6E+9 MOI의 작제물 SMSPV4 (n = 4), 3.6E+8 MOI의 작제물 SMSPV4 (n = 4), 3.6E+9 MOI의 작제물 SMSPV5 (n = 4), 3.6E+8 MOI의 작제물 SMSPV5 (n = 4), 3.6E+9 MOI의 작제물 SMSPV6 (n = 4), 그리고 3.6E+8 MOI의 작제물 SMSPV6 (n = 4)으로 처리 전 (열린 막대) 및 처리 후 (채워진 막대), 오픈 필드 상자에서 10 분의 자발적 탐색 동안 만성 뇌전증 생쥐에 의한 이동된 총 거리를 보여주는 막대 그래프이다. SMSPV4, SMSPV5 및 SMSPV6으로 생쥐의 처리는 생쥐에서, 뇌전증 행동에 대한 프록시인 과운동을 감소시키지만, 대조 작제물은 그렇지 않았다. 프리 = 처리전; 포스트 = 처리후; E8 = 3.6E+8 MOI; E9 = 3.6E+9 MOI.
상세한 설명
개체 (예컨대 포유류 개체, 예를 들면, 인간)에서 뇌전증, 예컨대 예를 들면, 측두엽 뇌전증 (TLE; 예를 들면, 치료에 불응성인 TLE)의 치료를 위한 조성물과 방법이 본원에서 설명된다. 예를 들면, 글루타민산염 이온성 수용체 카이네이트 유형 아단위 2 (Grik2) 유전자에 의해 인코딩된 mRNA를 표적으로 하는 저해성 RNA 분자 (예를 들면, 안티센스 올리고뉴클레오티드 (ASO) 또는 이것을 인코딩하는 핵산 벡터, 예컨대 본원에서 설명된 것들)의 치료 효과량이 예를 들면, 치료가 필요한 개체에서 뇌전증을 치료하기 위해 본원에서 설명된 방법에 따라서 투여될 수 있다. TLE의 치료를 위해 Grik2 mRNA를 표적으로 하는 ASO 작용제를 인코딩하는 핵산 벡터 (예를 들면, 바이러스 벡터, 예컨대 예를 들면, 렌티바이러스 또는 아데노 관련 바이러스 (AAV) 벡터)를 내포하는 조성물이 본원에서 설명된다.
Grik2
Grik2는 효현제 카이네이트에 의해 선택적으로 활성화될 수 있는 이온성 글루타민산염 수용체 아단위, GluK2를 인코딩하는 유전자이다. GluK2 내포 카이네이트 수용체 (KAR)는 다른 이온성 글루타민산염 수용체와 마찬가지로, 나트륨 및 칼륨에 투과성인 양이온 통로 포어를 개방하는 작용을 하는 글루타민산염에 의한 빠른 리간드 게이팅을 나타낸다. KAR 복합체는 여러 아단위로부터 KAR 아단위의 이종 또는 동종 조립체로서 조립될 수 있다. 이런 수용체는 리간드 결합 도메인을 함께 형성하는 세포외 N 말단 및 큰 펩티드 루프, 그리고 및 세포내 C 말단을 특징으로 한다. 이온성 글루타민산염 수용체 복합체는 그 자체로, 리간드-게이팅 이온 통로로서 작용하고, 그리고 글루타민산염에 결합 시에, 뉴런 막을 교차하여 하전된 이온의 통과를 매개한다. 일반적으로, KAR은 GluK1, 2 및/또는 3 (이전에는 각각, GluR5, GluR6 및 GluR7로 명명됨), GluK4 (KA1) 및 GluK5 (KA2) 아단위의 다중결합 조립체이다 (Collingridge, Neuropharmacology. 2009 Jan;56(1):2-5). KAR 복합체에 관련된 아단위의 다양한 조합은 종종, 특정 KAR 아단위를 인코딩하는 mRNA의 RNA 스플라이싱 및/또는 RNA 편집 (예를 들면, 아데노신 탈아미노효소에 의한 아데노신의 이노신으로의 전환)에 의해 결정된다. 게다가, 이런 RNA 변형은 상기 수용체의 특성에 영향을 줄 수 있다, 예컨대 예를 들면, 상기 통로의 칼슘 투과성을 변경할 수 있다. GluK2 내포 KAR은 이온성 글루타민산염 수용체 활성의 조정, 그리고 차후에, 뇌전증발생에 관련된 증상의 개선을 위한 적합한 표적이다.
측두엽 뇌전증
뇌전증발생은 뇌전증의 확립을 야기하고, 그리고 병리학적 뉴런 네트워크 재편성으로 이어지는 세포, 분자 및 형태학적 변화가 발생하는 동안 잠재하는 것으로 보일 수 있는 과정이다. TLE는 뇌전증유발 병소의 해부학적 기원에 근거된 2가지 주된 유형에 의해 특징화된다. 내측 측두엽 (예를 들면, 그 중에서도 특히, 해마, 해마방회, 서브이큘럼 및 편도)으로부터 유래하는 TLE는 내측 TLE (mTLE)로 명명되고, 반면 외측 측두엽 (예를 들면, 관자 신피질)으로부터 유래하는 TLE는 편측 TLE (lTLE)로서 지칭된다. TLE에 특유한 추가 특질은 해마의 CA1, CA3, 치아 문 및 치상회 (DG) 영역에서 뉴런 세포 사멸, GABA 역전 전위의 역전, DG에서 과립 세포 (GC) 분산, 그리고 치아 GC 위에 병리생리학적 재발성 흥분성 시냅스의 형성 (rMF-DGC 시냅스)을 야기하는 재발성 GC 이끼 섬유의 발아를 포함할 수 있다.
다양한 원인 인자가 내측 측두엽 경화증, 외상성 뇌 손상, 뇌 감염 (예를 들면, 뇌염 및 수막염), 저산소 뇌 손상, 뇌졸중, 대뇌 종양, 유전적 증후군, 그리고 열성 발작을 비롯한, TLE의 병인에 기인하였다. CNS의 가소성이 발달 상태 및 뇌 영역 특이적 감수성 둘 모두에 의존하기 때문에, 뇌 손상을 겪는 개체에서 반드시 뇌전증이 발달하는 것은 아니다. DG를 비롯한, 해마가 TLE를 야기하는 손상에 특히 감수성인 뇌 영역으로서 확인되었고, 그리고 일부 경우에, 치료 내성 (다시 말하면, 난치성) 뇌전증과 연관되었다 (Jarero-Basulto, J.J., et al. Pharmaceuticals, 2018, 11, 17; doi:10.3390/ph11010017). 흥분성 글루타민산성 신호전달의 증폭은 자연발생적 발작을 조장할 수 있다 (Kuruba, et al. Epilepsy Behav. 2009, 14 (Suppl. 1), 65-73). 화학적 글루타민산염 저해제, 예를 들면 NMDA 수용체 길항제는 글루타민산염 매개 흥분독성 및 급성 발작 생성에 의한 뉴런 사멸을 차단하거나 또는 감소시키는 것으로 나타났다. 하지만, 이런 작용제는 TLE에서 불량한 효능을 나타낸다 (Foster, AC, and Kemp, JA. Curr. Opin. Pharmacol. 2006, 6, 7-17).
이론에 한정됨 없이, 이소성 GluK2 내포 KAR을 통해 작동하는 일탈적 rMF-DGC 시냅스 (Epsztein et al., 2005; Artinian et al., 2011, 2015)는 TLE에서 만성 발작에서 핵심적인 역할을 수행할 수 있다 (Peret et al., 2014). 예를 들면, 발작간 극파 및 발작성 사건 (다시 말하면, 뇌전증모양 뇌 활성의 전기생리학적 시그너처)이 GluK2 수용체 아단위를 결여하는 유전자도입 생쥐에서, 또는 GluK2/GluK5 수용체를 저해하는 약리학적 작용제의 존재에서 감소되었다 (Peret et al., 2014; Crpel and Mulle, 2015). 이들 이론을 검정하기 위해 설계된 유전자도입 동물 모형에서 GluK2의 녹다운 또는 침묵이 실현가능하긴 하지만, GluK2 아단위에 대해 선택적이고 인간에게 이용하는 데 안전한 저해제를 설계하는 것은 난제이다. GluK 아단위는 구조적으로 보존되고, 그리고 이들의 DNA 코딩 서열은 유의미한 상동성을 공유한다. 동종체와 이종체 이온성과 대사성 글루타민산염 수용체에 대하여 뇌에서 복잡한 유전자 발현 패턴은 치료적 전략을 더욱 복잡하게 만든다. 본원에서 개시된 방법과 조성물은 Grik2 mRNA를 표적으로 하고 뉴런 또는 성상세포에서 GluK2 내포 KAR의 발현을 감소시키며 (예를 들면, 녹다운시키며), 그것에 의하여 예를 들면, 뉴런 회로 (예를 들면, 해마 회로)에서 자연발생적 뇌전증모양 방전의 감소를 증진함으로써, TLE (예를 들면, mTLE 또는 lTLE)의 치료에 적합하다. 따라서, 본원에서 설명된 조성물과 방법은 상기 질환의 생리학적 원인을 표적으로 하고 치유 요법에 이용될 수 있다.
Grik2
mRNA를 표적으로 하는 올리고뉴클레오티드 작용제
TLE의 임상적 관리는 극히 어려운데, TLE 환자 중 1/3이 가용한 약제를 이용하여 심신쇠약성 발작을 적절히 통제할 수 없다. 이들 환자는 종종, 치료에 불응성인 재발성 뇌전증 발작을 경험한다. 이런 시나리오에서, TLE 환자는 측두엽 내에 뇌전증유발 병소의 침습성 및 비가역성 외과적 적출에 의존할 수 있는데, 이것은 원치 않는 인지 결함을 야기할 수 있다. 따라서, TLE 환자 중 실제적인 분율이 약제 내성 TLE를 치료하기 위한 신규한 치료적 도정을 필요로 한다. 본원에서 설명된 조성물과 방법은 TLE의 발달과 진행을 야기하는 근원적인 분자 병태생리학을 치료하는 유익성을 제공한다.
Grik2 mRNA (예를 들면, 서열 번호: 115-125 중에서 하나)를 표적으로 하는 저해성 RNA 작제물 (예를 들면, ASO 작용제 또는 이들을 인코딩하는 핵산 벡터)을 인코딩하는 폴리뉴클레오티드인 본원에서 설명된 조성물이 TLE를 치료하기 위한 본원에서 설명된 방법에 따라서 투여될 수 있다. 본원에서 설명된 방법과 조성물은 임의의 유형의 TLE, 예컨대 예를 들면, 초점성 발작을 동반한 TLE, 전신 발작을 동반한 TLE, mTLE, 또는 lTLE를 겪는 TLE 환자를 치료하는 데 이용될 수 있다. 게다가, 본원에서 개시된 방법과 조성물은 임의의 병인 예컨대 예를 들면, 내측 측두엽 경화증, 외상성 뇌 손상, 뇌 감염 (예를 들면, 뇌염 및 수막염), 저산소 뇌 손상, 뇌졸중, 대뇌 종양, 유전적 증후군, 또는 열성 발작으로부터 발생하는 TLE를 치료하는 데 이용될 수 있다. 본원에서 설명된 조성물과 방법은 또한, TLE가 발달할 위험에 처해 있는 개체, 예를 들면, TLE 진행의 잠재기에 있는 개체에 대한 예방적 처치로서 투여될 수 있다.
본원에서 개시된 방법과 조성물에 따라서, ASO는 세포 (예를 들면, 뉴런, 예컨대 예를 들면, 해마 뉴런, 예컨대 예를 들면, 치상회, 예컨대 예를 들면, 치상 과립 세포 (DGC)의 해마 뉴런)에서 Grik2 mRNA의 분해를 야기하고, 그것에 의하여 상기 mRNA의 기능적 GluK2 단백질로의 번역을 예방함으로써 Grik2 mRNA의 발현을 저해할 수 있다.
본원에서 개시된 Grik2 mRNA를 표적으로 하는 ASO 작용제는 하나 이상의 뇌 영역에서 뇌전증 뇌 활성 (예를 들면, 뇌전증모양 방전)의 빈도를 감소시키거나 또는 이의 발생을 완전히 저해하는 작용을 할 수 있다. 이런 뇌 영역은 내측 측두엽, 외측 측두엽, 전두엽, 또는 더욱 구체적으로, 해마 (예를 들면, DG, CA1, CA2, CA3, 서브이큘럼) 또는 신피질을 포함할 수 있지만 이들에 한정되지 않는다. DG의 rMF-DGC에서 GluK2 내포 KAR의 일탈적 발현으로 인해, 뇌전증 뇌 활성의 발생이 DG에서 저해될 수 있다.
따라서, 본원 발명은 서열 번호: 1-100 중에서 하나에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO 또는 이것을 인코딩하는 핵산 벡터의 효과량을 CNS 세포 (예를 들면, DGC)와 접촉시킴으로써, 상기 세포에서 뇌전증모양 방전을 감소시키기 위한 방법과 조성물을 제공한다.
본원 발명의 ASO 작용제는 GluK2 저해제일 수 있다. 특히, GluK2 저해제는 Grik2 mRNA 발현 저해제일 수 있다. GluK2의 발현을 저해하는 것은 GluK5의 수준을 또한 저해할 수 있다 (Ruiz et al, J Neuroscience 2005). 임의의 이론에 한정됨 없이, 본원 발명은 GluK5 아단위 단독이 동종체 조립을 형성할 수 없기 때문에, GluK2 단독의 충분한 제거가 모든 GluK2/GluK5 이종체를 제거할 것이라는 원리에 기초된다.
개시된 방법과 조성물에 따라서, 본원에서 개시된 ASO 작용제는 15개 내지 50개 뉴클레오티드 (예를 들면, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 또는 25, 30, 35, 40, 45개, 또는 50개까지 뉴클레오티드)의 길이를 가질 수 있다. 예를 들면, 본원에서 개시된 ASO 작용제는 15개 뉴클레오티드의 길이를 가질 수 있다. 다른 실례에서, ASO 작용제는 16개 뉴클레오티드의 길이를 갖는다. 다른 실례에서, ASO 작용제는 17개 뉴클레오티드의 길이를 갖는다. 다른 실례에서, ASO 작용제는 18개 뉴클레오티드의 길이를 갖는다. 다른 실례에서, ASO 작용제는 19개 뉴클레오티드의 길이를 갖는다. 다른 실례에서, ASO 작용제는 20개 뉴클레오티드의 길이를 갖는다. 다른 실례에서, ASO 작용제는 21개 뉴클레오티드의 길이를 갖는다. 다른 실례에서, ASO 작용제는 22개 뉴클레오티드의 길이를 갖는다. 다른 실례에서, ASO 작용제는 23개 뉴클레오티드의 길이를 갖는다. 다른 실례에서, ASO 작용제는 24개 뉴클레오티드의 길이를 갖는다. 다른 실례에서, ASO 작용제는 25개 뉴클레오티드의 길이를 갖는다. 다른 실례에서, ASO 작용제는 25-30개 뉴클레오티드의 길이를 갖는다. 다른 실례에서, ASO 작용제는 30-35개 뉴클레오티드의 길이를 갖는다. 다른 실례에서, ASO 작용제는 35-40개 뉴클레오티드의 길이를 갖는다. 다른 실례에서, ASO 작용제는 40-45개 뉴클레오티드의 길이를 갖는다. 다른 실례에서, ASO 작용제는 45-50개 뉴클레오티드의 길이를 갖는다.
본원 발명의 ASO 작용제는 Grik2 mRNA의 서열 (예를 들면, 서열 번호: 115-689 중에서 하나) 또는 이의 변이체의 영역에 적어도 실제적으로 상보적이거나 또는 완전히 상보적인 서열을 포함하고, 상기 상보성은 세포내 조건하에 특이적 결합을 생성하는 데 충분하다. 예를 들면, 본원 발명은 Grik2 mRNA의 하나 이상의 영역의 적어도 7개 (예를 들면, 적어도 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22개, 또는 그 초과)의 연속 뉴클레오티드에 상보적인 안티센스 서열을 갖는 ASO 작용제를 예기한다. 특정 실례에서, ASO 작용제는 Grik2 mRNA의 하나 이상의 영역의 7개의 연속 뉴클레오티드에 상보적인 안티센스 서열을 갖는다. 다른 실례에서, ASO 작용제는 Grik2 mRNA의 하나 이상의 영역의 8개의 연속 뉴클레오티드에 상보적인 안티센스 서열을 갖는다. 다른 실례에서, ASO 작용제는 Grik2 mRNA의 하나 이상의 영역의 9개의 연속 뉴클레오티드에 상보적인 안티센스 서열을 갖는다. 다른 실례에서, ASO 작용제는 Grik2 mRNA의 하나 이상의 영역의 10개의 연속 뉴클레오티드에 상보적인 안티센스 서열을 갖는다. 다른 실례에서, ASO 작용제는 Grik2 mRNA의 하나 이상의 영역의 11개의 연속 뉴클레오티드에 상보적인 안티센스 서열을 갖는다. 다른 실례에서, ASO 작용제는 Grik2 mRNA의 하나 이상의 영역의 12개의 연속 뉴클레오티드에 상보적인 안티센스 서열을 갖는다. 다른 실례에서, ASO 작용제는 Grik2 mRNA의 하나 이상의 영역의 13개의 연속 뉴클레오티드에 상보적인 안티센스 서열을 갖는다. 다른 실례에서, ASO 작용제는 Grik2 mRNA의 하나 이상의 영역의 14개의 연속 뉴클레오티드에 상보적인 안티센스 서열을 갖는다. 다른 실례에서, ASO 작용제는 Grik2 mRNA의 하나 이상의 영역의 15개의 연속 뉴클레오티드에 상보적인 안티센스 서열을 갖는다. 다른 실례에서, ASO 작용제는 Grik2 mRNA의 하나 이상의 영역의 16개의 연속 뉴클레오티드에 상보적인 안티센스 서열을 갖는다. 다른 실례에서, ASO 작용제는 Grik2 mRNA의 하나 이상의 영역의 17개의 연속 뉴클레오티드에 상보적인 안티센스 서열을 갖는다. 다른 실례에서, ASO 작용제는 Grik2 mRNA의 하나 이상의 영역의 18개의 연속 뉴클레오티드에 상보적인 안티센스 서열을 갖는다. 다른 실례에서, ASO 작용제는 Grik2 mRNA의 하나 이상의 영역의 19개의 연속 뉴클레오티드에 상보적인 안티센스 서열을 갖는다. 다른 실례에서, ASO 작용제는 Grik2 mRNA의 하나 이상의 영역의 20개의 연속 뉴클레오티드에 상보적인 안티센스 서열을 갖는다. 다른 실례에서, ASO 작용제는 Grik2 mRNA의 하나 이상의 영역의 21개의 연속 뉴클레오티드에 상보적인 안티센스 서열을 갖는다. 다른 실례에서, ASO 작용제는 Grik2 mRNA의 하나 이상의 영역의 22개의 연속 뉴클레오티드에 상보적인 안티센스 서열을 갖는다. 또 다른 실례에서, ASO 작용제는 Grik2 mRNA의 하나 이상의 영역의 뉴클레오티드에 100% 상보적인 안티센스 서열을 갖는다.
본원 발명은 Grik2 mRNA의 하나 이상의 영역 (예를 들면, 서열 번호: 115-681에서 설명된 Grik2 mRNA의 영역 중에서 하나)에 결합될 때, 길이에서 7-22개 (예를 들면, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 또는 22개) 뉴클레오티드 사이의 Grik2 mRNA와의 이중나선 구조를 형성하는 ASO 작용제를 예기한다. 예를 들면, ASO 작용제 및 Grik2 mRNA 사이의 이중나선 구조는 길이에서 7개 뉴클레오티드일 수 있다. 다른 실례에서, ASO 작용제 및 Grik2 mRNA 사이의 이중나선 구조는 길이에서 8개 뉴클레오티드일 수 있다. 다른 실례에서, ASO 작용제 및 Grik2 mRNA 사이의 이중나선 구조는 길이에서 9개 뉴클레오티드일 수 있다. 다른 실례에서, ASO 작용제 및 Grik2 mRNA 사이의 이중나선 구조는 길이에서 10개 뉴클레오티드일 수 있다. 다른 실례에서, ASO 작용제 및 Grik2 mRNA 사이의 이중나선 구조는 길이에서 11개 뉴클레오티드일 수 있다. 다른 실례에서, ASO 작용제 및 Grik2 mRNA 사이의 이중나선 구조는 길이에서 12개 뉴클레오티드일 수 있다. 다른 실례에서, ASO 작용제 및 Grik2 mRNA 사이의 이중나선 구조는 길이에서 13개 뉴클레오티드일 수 있다. 다른 실례에서, ASO 작용제 및 Grik2 mRNA 사이의 이중나선 구조는 길이에서 14개 뉴클레오티드일 수 있다. 다른 실례에서, ASO 작용제 및 Grik2 mRNA 사이의 이중나선 구조는 길이에서 15개 뉴클레오티드일 수 있다. 다른 실례에서, ASO 작용제 및 Grik2 mRNA 사이의 이중나선 구조는 길이에서 16개 뉴클레오티드일 수 있다. 다른 실례에서, ASO 작용제 및 Grik2 mRNA 사이의 이중나선 구조는 길이에서 17개 뉴클레오티드일 수 있다. 다른 실례에서, ASO 작용제 및 Grik2 mRNA 사이의 이중나선 구조는 길이에서 18개 뉴클레오티드일 수 있다. 다른 실례에서, ASO 작용제 및 Grik2 mRNA 사이의 이중나선 구조는 길이에서 19개 뉴클레오티드일 수 있다. 다른 실례에서, ASO 작용제 및 Grik2 mRNA 사이의 이중나선 구조는 길이에서 20개 뉴클레오티드일 수 있다. 다른 실례에서, ASO 작용제 및 Grik2 mRNA 사이의 이중나선 구조는 길이에서 21개 뉴클레오티드일 수 있다. 또 다른 실례에서, ASO 작용제 및 Grik2 mRNA 사이의 이중나선 구조는 길이에서 10개 뉴클레오티드일 수 있다.
개시된 방법과 조성물에 따라서, ASO 작용제 (예를 들면, 본원에서 개시된 ASO 작용제 중에서 하나, 예컨대 예를 들면, 서열 번호: 1-100의 ASO 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체 중에서 하나), 그리고 Grik2 mRNA의 하나 이상의 영역에 의해 형성된 이중나선 구조는 적어도 하나 (예를 들면, 적어도 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 또는 15개)의 부정합을 포함할 수 있다. 예를 들면, 이중나선 구조는 1개의 부정합을 내포할 수 있다. 다른 실례에서, 이중나선 구조는 2개의 부정합을 내포한다. 다른 실례에서, 이중나선 구조는 3개의 부정합을 내포한다. 다른 실례에서, 이중나선 구조는 4개의 부정합을 내포한다. 다른 실례에서, 이중나선 구조는 5개의 부정합을 내포한다. 다른 실례에서, 이중나선 구조는 6개의 부정합을 내포한다. 다른 실례에서, 이중나선 구조는 7개의 부정합을 내포한다. 다른 실례에서, 이중나선 구조는 8개의 부정합을 내포한다. 다른 실례에서, 이중나선 구조는 9개의 부정합을 내포한다. 다른 실례에서, 이중나선 구조는 10개의 부정합을 내포한다. 다른 실례에서, 이중나선 구조는 11개의 부정합을 내포한다. 다른 실례에서, 이중나선 구조는 12개의 부정합을 내포한다. 다른 실례에서, 이중나선 구조는 13개의 부정합을 내포한다. 다른 실례에서, 이중나선 구조는 14개의 부정합을 내포한다. 또 다른 실례에서, 이중나선 구조는 15개의 부정합을 내포한다.
따라서, 본원 발명의 목적은 Grik2 mRNA를 표적으로 하는 단리된, 합성, 또는 재조합 ASO 작용제에 관계한다. 본원 발명의 ASO 작용제는 RNA 또는 DNA 올리고뉴클레오티드를 비롯한, 임의의 적합한 유형일 수 있다. 따라서, 개시된 방법과 조성물은 ASO 작용제 (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA, 또는 shmiRNA)인 Grik2 발현 저해제를 특징으로 한다. 안티센스 RNA 분자 및 안티센스 DNA 분자를 비롯한, ASO 작용제는 Grik2 mRNA에 결합하고 단백질 번역을 예방하거나 또는 mRNA 분해를 증가시킴으로써 이것의 번역을 직접적으로 차단하고, 그것에 의하여 GluK2 단백질의 수준과 활성을 감소시키는 작용을 할 수 있다. 예를 들면, 적어도 약 19개의 염기 및 GluK2를 인코딩하는 mRNA 전사체 서열의 고유한 영역에 상보성을 갖는 ASO 작용제가 예를 들면, 전통적인 기술 (예를 들면, 본원에서 개시된 기술)에 의해 합성되고, 그리고 예를 들면, 본원에서 설명된 다른 루트 중에서 정맥내 주사 또는 주입, 예컨대 뇌의 영역에 직접 주사에 의해 투여될 수 있다. 서열이 알려져 있는 유전자의 유전자 발현을 특이적으로 경감하기 위해 안티센스 기술을 이용하기 위한 방법은 당해 분야에서 널리 알려져 있다 (참조: 예를 들면 U.S. 특허 번호 6,566,135; 6,566,131; 6,365,354; 6,410,323; 6,107,091; 6,046,321; 및 5,981,732, 이들은 각각 본원에서 온전히 참조로서 편입된다).
특정 실례에서, 본원 발명의 Grik2 ASO 작용제는 짧은 간섭 RNA (siRNA)일 수 있다. Grik2 유전자 발현은 개체 또는 세포를 작은 이중 가닥 RNA (dsRNA), 또는 이것을 인코딩하는 벡터와 접촉시키고, 그것에 의하여 서열 특이적 방식으로 (예를 들면, RNA 간섭 경로에 의하여) mRNA의 분해에 의해 Grik2 발현을 특이적으로 저해할 수 있는 작은 이중 가닥 RNA의 생산을 야기함으로써 감소될 수 있다. 서열이 알려져 있는 유전자에 대한 적합한 dsRNA 또는 dsRNA-인코딩 벡터를 선택하기 위한 방법은 당해 분야에서 알려져 있다 (참조: 예를 들면 Tuschl, T. et al. (1999); Elbashir, S. M. et al. (2001); Hannon, GJ. (2002); McManus, MT. et al. (2002); Brummelkamp, TR. et al. (2002); U.S. 특허 번호 6,573,099 및 6,506,559; 그리고 국제 특허 공개 번호 WO 01/36646, WO 99/32619 및 WO 01/68836, 이들은 각각 본원에서 온전히 참조로서 편입된다).
본원 발명의 Grik2 ASO 작용제는 또한, 짧은 헤어핀 RNA (shRNA)일 수 있다. shRNA는 RNA 간섭을 통해 유전자 발현을 침묵시키는 데 이용될 수 있는 단단한 헤어핀 회전을 만드는 RNA의 서열이다. shRNA는 일반적으로, 표적 세포 내로 도입된 벡터를 이용하여 발현되는데, 여기서 상기 벡터는 shRNA가 구조성으로 발현되도록 담보하기 위해 편재성 U6 프로모터를 종종 활용한다. 이러한 벡터는 통상적으로, 딸세포에게 전달되어, 세포 분열 이후에 유전자 침묵이 유지될 수 있도록 한다. shRNA 헤어핀 구조는 세포 기구에 의해 siRNA로 개열되고, 이것은 이후, RNA 유발 침묵 복합체 (RISC)에 결합된다. 이러한 복합체는 mRNA에 결합하고 이들을 개열하는데, 이들은 자신이 결합되는 siRNA 서열과 정합한다.
추가적으로, 본원 발명의 Grik2 발현 저해제는 마이크로RNA (miRNA)일 수 있다. miRNA는 당해 분야에서의 일반적인 의미를 갖고, 그리고 예를 들면, 비록 19개 및 23개까지 뉴클레오티드의 길이가 보고되긴 했지만 일반적으로 길이에서 21개 내지 22개 뉴클레오티드이고 표적화된 mRNA의 번역을 억제하는 데 이용될 수 있는 마이크로RNA 분자를 지칭한다. miRNA는 각각, 더 긴 전구체 RNA 분자 ("전구체 miRNA")로부터 처리된다. 전구체 miRNA는 비단백질-인코딩 유전자로부터 전사된다. 전구체 miRNA는 그들이 스템 루프 또는 폴드 백 유사 구조를 형성할 수 있게 하는 상보성의 두 영역을 갖는데, 상기 구조는 동물에서 Dicer로 불리는 리보핵산분해효소 III 유사 뉴클레아제 효소에 의해 개열된다. 처리된 miRNA는 전형적으로, 표적 mRNA의 영역에 완전히 또는 실제적으로 상보적인 "시드 서열" (전형적으로 6-8개 뉴클레오티드)을 내포하는 스템의 일부이다. 처리된 miRNA ("성숙 miRNA"로서 또한 지칭됨)는 특정 표적 유전자를 하향조절하는 (예를 들면, miRNA의 번역을 감소시키거나 또는 이것을 분해하는) 큰 복합체의 일부가 된다.
게다가, 본원 발명의 GluK2 저해제는 miRNA 적응 shRNA (shmiRNA)이다. shmiRNA 작용제는 마이크로RNA 측접 및 루프 서열을 내포하는 마이크로RNA 골격 (예를 들면, miR-30 골격)의 -5p 또는 -3p 팔 내에 안티센스 서열을 통합하는 키메라 분자를 지칭한다. shRNA와 비교하여, shmiRNA는 일반적으로, 마이크로RNA 유래된 서열에 기초된 더 긴 스템 루프 구조를 갖는데, -5p 및 -3p 팔이 완전한 또는 실제적인 상보성 (예를 들면, 부정합, G:U 동요)을 나타낸다. 더 긴 서열 및 처리 요건으로 인해, shmiRNA는 일반적으로, Pol II 프로모터로부터 발현된다. 이들 작제물은 또한, shRNA 기반 작용제와 비교하여 감소된 독성을 나타내는 것으로 밝혀졌다.
복수의 miRNA가 Grik2 mRNA 발현 (및 차후에 이의 유전자 산물, GluK2)을 녹다운시키는 데 이용될 수 있다. 이들 miRNA는 상이한 표적 전사체 또는 단일 표적 전사체의 상이한 결합 부위에 상보적일 수 있다. 다중유전자 또는 다중유전자 전사체가 또한, 표적 유전자 녹다운의 효율을 증강하는 데 활용될 수 있다. 동일한 miRNA 또는 상이한 miRNA를 인코딩하는 복수의 유전자가 단일 전사체에서 함께, 또는 단일 벡터 카세트에서 별개의 전사체로서 조절될 수 있다. 본원 발명의 miRNA는 벡터, 예컨대 예를 들면, 재조합 아데노 관련 바이러스 (rAAV) 벡터, 렌티바이러스 벡터, 레트로바이러스 벡터 및 레트로트랜스포손 기반 벡터 시스템을 포함하지만 이들에 한정되지 않는 바이러스 벡터 내로 포장될 수 있다.
Grik2 mRNA의 센스 표적 서열에 상보적 (예를 들면, 실제적으로 또는 완전히 상보적인) ASO는 일반적으로, 임의의 전술한 저해제 (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA)의 생산을 위한 DNA 서열에 의해 인코딩된다. 관심되는 이중 가닥 RNA를 인코딩하는 DNA는 유전자 카세트 (예를 들면, DNA의 전사가 프로모터에 의해 제어되는 발현 카세트) 내로 통합될 수 있다.
안티센스 올리고뉴클레오티드 서열
본원 발명의 방법과 조성물에 따라서, 본원에서 개시된 저해성 RNA 작용제는 표 2에서 개시된 ASO 작용제 (예를 들면, 서열 번호: 1-100), 또는 아래에 나타나 있는 바와 같이, 서열 번호: 1-100 중에서 하나의 상응하는 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체 중에서 하나 이상을 포함할 수 있다. ASO 작용제는 아래의 표 4에서 설명된 Grik2 mRNA의 상응하는 표적 서열, 또는 서열 번호: 164-681 중에서 하나, 또는 아래의 표 4에서 설명된 상응하는 표적 서열 또는 서열 번호: 164-681 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에 결합할 수 있다.
표 2:
Grik2
mRNA를 표적으로 하는 안티센스 올리고뉴클레오티드 서열
주의: % KD 값은 실시예 1A에서 상세하게 설명된 바와 같이, 이중-루시페라아제 리포터 검정에서 상기 확인된 ASO 작용제에 의해 달성된 GluK2 단백질의 녹다운 퍼센트에 상응한다.
앞서 말한 서열은 본원 발명의 벡터 내로 통합될 수 있는 DNA (다시 말하면, cDNA) 서열로서 나타내진다; 하지만, 이들 서열은 또한, 세포 내에 벡터로부터 합성되는 상응하는 RNA 서열로서 나타내질 수 있다. 당업자는 cDNA 서열이 우리딘의 티미딘으로의 치환을 제외하고, mRNA 서열과 동등하고, 그리고 본원에서 동일한 목적, 다시 말하면, Grik2 mRNA의 발현을 저해하기 위한 안티센스 올리고뉴클레오티드의 생성에 이용될 수 있다는 것을 이해할 것이다. DNA 벡터 (예를 들면, AAV)의 경우에, 안티센스 핵산을 내포하는 폴리뉴클레오티드는 DNA 서열이다. RNA 벡터의 경우에, 도입유전자 카세트는 본원에서 설명된 안티센스 DNA 서열의 RNA 등가물을 통합한다.
본원 발명의 ASO 서열은 서열 번호: 1의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 1의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 1의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 1의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 2의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 2의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 2의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 2의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 3의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 3의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 3의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 3의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 4의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 4의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 4의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 4의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 5의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 5의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 5의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 5의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 6의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 6의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 6의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 6의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 7의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 7의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 7의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 7의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 8의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 8의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 8의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 8의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 9의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 9의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 9의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 9의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 10의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 10의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 10의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 10의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 11의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 11의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 11의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 11의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 12의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 12의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 12의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 12의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 13의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 13의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 13의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 13의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 14의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 14의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 14의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 14의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 15의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 15의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 15의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 15의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 16의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 16의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 16의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 16의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 17의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 17의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 17의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 17의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 18의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 18의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 18의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 18의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 19의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 19의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 19의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 19의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 20의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 20의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 20의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 20의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 21의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 21의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 21의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 21의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 22의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 22의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 22의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 22의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 23의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 23의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 23의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 23의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 24의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 24의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 24의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 24의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 25의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 25의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 25의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 25의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 26의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 26의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 26의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 26의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 27의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 27의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 27의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 27의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 28의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 28의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 28의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 28의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 29의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 29의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 29의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 29의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 30의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 30의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 30의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 30의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 31의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 31의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 31의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 31의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 32의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 32의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 32의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 32의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 33의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 33의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 33의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 33의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 34의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 34의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 34의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 34의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 35의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 35의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 35의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 35의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 36의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 36의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 36의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 36의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 37의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 37의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 37의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 37의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 38의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 38의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 38의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 38의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 39의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 39의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 39의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 39의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 40의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 40의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 40의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 40의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 41의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 41의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 41의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 41의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 42의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 42의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 42의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 42의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 43의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 43의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 43의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 43의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 44의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 44의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 44의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 44의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 45의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 45의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 45의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 45의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 46의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 46의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 46의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 46의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 47의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 47의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 47의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 47의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 48의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 48의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 48의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 48의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 49의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 49의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 49의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 49의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 50의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 50의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 50의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 50의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 51의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 51의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 51의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 51의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 52의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 52의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 52의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 52의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 53의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 53의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 53의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 53의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 54의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 54의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 54의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 54의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 55의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 55의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 55의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 55의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 56의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 56의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 56의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 56의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 57의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 57의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 57의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 57의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 58의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 58의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 58의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 58의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 59의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 59의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 59의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 59의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 60의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 60의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 60의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 60의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 61의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 61의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 61의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 61의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 62의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 62의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 62의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 62의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 63의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 63의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 63의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 63의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 64의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 64의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 64의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 64의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 65의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 65의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 65의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 65의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 66의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 66의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 66의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 66의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 67의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 67의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 67의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 67의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 68의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 68의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 68의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 68의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 69의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 69의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 69의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 69의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 70의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 70의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 70의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 70의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 71의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 71의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 71의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 71의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 72의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 72의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 72의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 72의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 73의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 73의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 73의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 73의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 74의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 74의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 74의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 74의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 75의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 75의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 75의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 75의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 76의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 76의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 76의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 76의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 77의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 77의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 77의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 77의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 78의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 78의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 78의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 78의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 79의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 79의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 79의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 79의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 80의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 80의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 80의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 80의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 81의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 81의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 81의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 81의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 82의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 82의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 82의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 82의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 83의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 83의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 83의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 83의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 84의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 84의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 84의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 84의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 85의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 85의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 85의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 85의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 86의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 86의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 86의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 86의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 87의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 87의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 87의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 87의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 88의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 88의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 88의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 88의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 89의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 89의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 89의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 89의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 90의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 90의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 90의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 90의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 91의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 91의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 91의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 91의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 92의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 92의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 92의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 92의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 93의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 93의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 93의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 93의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 94의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 94의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 94의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 94의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 95의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 95의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 95의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 95의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 96의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 96의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 96의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 96의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 97의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 97의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 97의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 97의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 98의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 98의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 98의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 98의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 99의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 99의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 99의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 99의 핵산 서열을 가질 수 있다.
본원 발명의 ASO 서열은 서열 번호: 100의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 예를 들면, ASO는 서열 번호: 100의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 다른 실례에서, ASO는 서열 번호: 100의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있다. 추가 실례에서, ASO는 서열 번호: 100의 핵산 서열을 가질 수 있다.
동요 염기쌍을 갖는 안티센스 올리고뉴클레오티드
본원 발명은 하나 이상의 동요 염기쌍을 갖는 ASO 작용제를 더욱 특징으로 한다. 4개의 주된 동요 염기쌍은 구아닌-우라실 (G-U), 히포크산틴-우라실 (I-U), 히포크산틴-아데닌 (I-A) 및 히포크산틴-시토신 (I-C)인데, 여기서 히포크산틴은 뉴클레오시드 이노신을 대표한다. G-U 동요 염기쌍은 G-C, A-T 및 A-U의 것과 유사한 열역학적 안정성을 나타내는 것으로 밝혀졌다 (Saxena et al, 2003, J Biol Chem, 278(45):44312-9).
따라서, 본원 발명은 서열 번호: 115 또는 서열 번호: 116의 표적 영역의 보체에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 뉴클레오티드 서열을 갖는 ASO 작용제를 제공한다 (예를 들면, ASO는 Grik2 유전자 서열의 안티센스 가닥에 적어도 85% 서열 동일성을 가질 수 있다). 특히, 본원 발명의 ASO 작용제는 상응하는 정렬된 인간 Grik2 mRNA 전사체 (예를 들면, 서열 번호: 115 또는 서열 번호: 116)에 상보적이지 않는 1, 2 또는 3개의 뉴클레오티드를 가질 수 있다. 따라서, 본원 발명의 ASO 작용제는 서열 번호: 116 또는 서열 번호: 115의 표적 영역의 보체에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과), 적어도 86% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과), 적어도 87% (예를 들면, 적어도 87%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과), 적어도 88% (예를 들면, 적어도 88%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과), 적어도 89% (예를 들면, 적어도 89%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과), 또는 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과) 동일한 뉴클레오티드 서열을 가질 수 있다. 정렬된 Grik2 mRNA 서열의 상보성 서열과 100% 동일하지 않는 뉴클레오티드는 동요 뉴클레오티드일 수 있다.
본원의 표 2에 나타나 있는 바와 같이, 5' 단부에서 소문자 'u'를 갖는 ASO 작용제는 표 2에서 열거된 다른 인간 ASO 작용제에 비하여, 인간 Grik2 mRNA의 상보성 서열과 동일한 하나 적은 뉴클레오티드를 갖는다. 5' 단부에서 'u'의 포함 (G:U 동요 염기쌍을 야기)은 RISC 부하를 향상시키기 위해 실행되었다 (siSPOTR 소프트웨어, Boudreau, R.L. et al., Nucleic Acid Res 2013, 41(1):e9).
Grik2 전사체 상에서 특정 영역에 대하여 설계된 안티센스 RNA에 의해 매개된 표적 이탈 효과의 확률은 수많은 공개적으로 가용한 알고리즘을 이용하여 계측될 수 있다. 예를 들면, 온라인 도구 siSPOTR (world-web.sispotr.icts.uiowa.edu/sispotr/index.html_에서 가용한 "siRNA Sequence Probability-of-Off-Targeting Reduction"이 이용될 수 있다).
일정한 Grik2 안티센스 서열은 "특이적" siSPOTR 가이드인 것으로 결정되었고 (표적 이탈 예측자 프로그램 siSPOTR에 근거하여), 그리고 서열 번호: 115 또는 서열 번호: 116을 비롯한 전사체의 서열 특이적 저해를 유지하면서 인간 유전체에서 표적 이탈 서열 특이적 유전자 억제를 방지하거나 또는 감소시킬 것으로 예측된 안티센스 RNA이다 (표 3 참조).
일정한 Grik2 안티센스 RNA는 "공유된" siSPOTR 서열인 것으로 결정되었고 (표적 이탈 예측자 프로그램 siSPOTR에 근거하여), 그리고 인간 유전체에서 표적 이탈 서열 특이적 유전자 억제를 방지하거나 또는 감소시키고 인간, 원숭이 및 생쥐 Grik2 mRNA 서열 사이에 유의미한 공유된 상동성을 가질 것으로 예측되었고 서열 번호: 115, 서열 번호: 116 (뿐만 아니라 서열 번호: 117-125)을 비롯한 전사체의 서열 특이적 저해를 유지할 것으로 예상되는 안티센스 RNA이다.
표 3: siSPOTR 예측된 Grik2 mRNA 안티센스 서열
본원에서 개시된 ASO 작용제는 GluK2 단백질 (예를 들면, 서열 번호: 102-114 중에서 하나를 포함하는 GluK2 단백질, 또는 서열 번호: 102의 적어도 아미노산 1 내지 509를 포함하는 GluK2 단백질)을 인코딩하는 mRNA를 표적으로 한다. GluK2 단백질을 인코딩하는 mRNA는 서열 번호: 102-114 중에서 하나의 서열을 갖는 폴리펩티드에 비하여, 하나 이상의 아미노산 치환, 예컨대 하나 이상의 보존성 아미노산 치환 (예를 들면, 1, 2, 3, 4, 5, 6, 7, 8, 9, 또는 10개 또는 그 초과의 아미노산 치환, 예컨대 1, 2, 3, 4, 5, 6, 7, 8, 9, 또는 10개 또는 그 초과의 보존성 아미노산 치환)을 내포하는 폴리펩티드를 인코딩하는 폴리뉴클레오티드를 포함할 수 있다.
본원에서 개시된 Grik2 ASO 작용제는 예를 들면, 생물정보학적 도구를 이용하여, Grik2 mRNA의 서열을 시작점으로서 이용함으로써 설계될 수 있다. Grik2 mRNA 서열은 NCBI 유전자 식별 번호: 2898에서 발견될 수 있다. 다른 실례에서, 서열 번호: 102를 인코딩하는 폴리뉴클레오티드 서열, 서열 번호: 102의 연속 아미노산 1 내지 509를 인코딩하는 폴리뉴클레오티드 서열, 또는 서열 번호: 102 (UniProtKB Q13002-1), 서열 번호: 103 (UniProtKB Q13002-2), 서열 번호: 104 (UniProtKB Q13002-3), 서열 번호: 105 (UniProtKB Q13002-4), 서열 번호: 106 (UniProtKB Q13002-5), 서열 번호: 107 (UniProtKB Q13002-6), 서열 번호: 108 (UniProtKB Q13002-7), 서열 번호: 109 (NCBI 수탁 번호: NP_001104738.2), 서열 번호: 110 (NCBI 수탁 번호: NP_034479.3), 서열 번호: 111 (NCBI 수탁 번호: NP_034479.3), 서열 번호: 112 (NCBI 수탁 번호: XP_014992481.1), 서열 번호: 113 (NCBI 수탁 번호: XP_014992483.1) 및 서열 번호: 114 (NCBI 수탁 번호: NP_062182.1) 중에서 하나의 아미노산 서열을 인코딩하는 폴리뉴클레오티드 서열이 GluK2 단백질을 인코딩하는 mRNA를 표적으로 하는 핵산을 설계하기 위한 기초로서 이용될 수 있다. GluK2 수용체를 인코딩하는 폴리뉴클레오티드 서열은 서열 번호: 115-125 중 하나에서 선택될 수 있다.
GluK2 폴리펩티드는 서열 번호: 102의 아미노산 서열을 가질 수 있거나 또는 서열 번호: 102의 아미노산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있는데, 이것은 아래에 도시된다 (UniProt Q13002-1; GRIK2_HUMAN 글루타민산염 수용체 이온성, 카이네이트 2):
MKIIFPILSNPVFRRTVKLLLCLLWIGYSQGTTHVLRFGGIFEYVESGPMGAEELAFRFA
VNTINRNRTLLPNTTLTYDTQKINLYDSFEASKKACDQLSLGVAAIFGPSHSSSANAVQS
ICNALGVPHIQTRWKHQVSDNKDSFYVSLYPDFSSLSRAILDLVQFFKWKTVTVVYDDST
GLIRLQELIKAPSRYNLRLKIRQLPADTKDAKPLLKEMKRGKEFHVIFDCSHEMAAGILK
QALAMGMMTEYYHYIFTTLDLFALDVEPYRYSGVNMTGFRILNTENTQVSSIIEKWSMER
LQAPPKPDSGLLDGFMTTDAALMYDAVHVVSVAVQQFPQMTVSSLQCNRHKPWRFGTRFM
SLIKEAHWEGLTGRITFNKTNGLRTDFDLDVISLKEEGLEKIGTWDPASGLNMTESQKGK
PANITDSLSNRSLIVTTILEEPYVLFKKSDKPLYGNDRFEGYCIDLLRELSTILGFTYEI
RLVEDGKYGAQDDANGQWNGMVRELIDHKADLAVAPLAITYVREKVIDFSKPFMTLGISI
LYRKPNGTNPGVFSFLNPLSPDIWMYILLAYLGVSCVLFVIARFSPYEWYNPHPCNPDSD
VVENNFTLLNSFWFGVGALMQQGSELMPKALSTRIVGGIWWFFTLIIISSYTANLAAFLT
VERMESPIDSADDLAKQTKIEYGAVEDGATMTFFKKSKISTYDKMWAFMSSRRQSVLVKS
NEEGIQRVLTSDYAFLMESTTIEFVTQRNCNLTQIGGLIDSKGYGVGTPMGSPYRDKITI
AILQLQEEGKLHMMKEKWWRGNGCPEEESKEASALGVQNIGGIFIVLAAGLVLSVFVAVG
EFLYKSKKNAQLEKRSFCSAMVEELRMSLKCQRRLKHKPQAPVIVKTEEVINMHTFNDRR
LPGKETMA
(서열 번호: 102)
GluK2 폴리펩티드는 서열 번호: 103의 아미노산 서열을 가질 수 있거나 또는 서열 번호: 103의 아미노산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있는데, 이것은 아래에 도시된다 (UniProt Q13002-2; 글루타민산염 수용체 이온성, 카이네이트 2의 GRIK2_HUMAN 동종형 2):
MKIIFPILSNPVFRRTVKLLLCLLWIGYSQGTTHVLRFGGIFEYVESGPMGAEELAFRFA
VNTINRNRTLLPNTTLTYDTQKINLYDSFEASKKACDQLSLGVAAIFGPSHSSSANAVQS
ICNALGVPHIQTRWKHQVSDNKDSFYVSLYPDFSSLSRAILDLVQFFKWKTVTVVYDDST
GLIRLQELIKAPSRYNLRLKIRQLPADTKDAKPLLKEMKRGKEFHVIFDCSHEMAAGILK
QALAMGMMTEYYHYIFTTLDLFALDVEPYRYSGVNMTGFRILNTENTQVSSIIEKWSMER
LQAPPKPDSGLLDGFMTTDAALMYDAVHVVSVAVQQFPQMTVSSLQCNRHKPWRFGTRFM
SLIKEAHWEGLTGRITFNKTNGLRTDFDLDVISLKEEGLEKIGTWDPASGLNMTESQKGK
PANITDSLSNRSLIVTTILEEPYVLFKKSDKPLYGNDRFEGYCIDLLRELSTILGFTYEI
RLVEDGKYGAQDDANGQWNGMVRELIDHKADLAVAPLAITYVREKVIDFSKPFMTLGISI
LYRKPNGTNPGVFSFLNPLSPDIWMYILLAYLGVSCVLFVIARFSPYEWYNPHPCNPDSD
VVENNFTLLNSFWFGVGALMQQGSELMPKALSTRIVGGIWWFFTLIIISSYTANLAAFLT
VERMESPIDSADDLAKQTKIEYGAVEDGATMTFFKKSKISTYDKMWAFMSSRRQSVLVKS
NEEGIQRVLTSDYAFLMESTTIEFVTQRNCNLTQIGGLIDSKGYGVGTPMGSPYRDKITI
AILQLQEEGKLHMMKEKWWRGNGCPEEESKEASALGVQNIGGIFIVLAAGLVLSVFVAVG
EFLYKSKKNAQLEKESSIWLVPPYHPDTV
(서열 번호: 103)
GluK2 폴리펩티드는 서열 번호: 104의 아미노산 서열을 가질 수 있거나 또는 서열 번호: 104의 아미노산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있는데, 이것은 아래에 도시된다 (UniProt Q13002-3; 글루타민산염 수용체 이온성, 카이네이트 2의 GRIK2_HUMAN 동종형 3):
MKIIFPILSNPVFRRTVKLLLCLLWIGYSQGTTHVLRFGGIFEYVESGPMGAEELAFRFA
VNTINRNRTLLPNTTLTYDTQKINLYDSFEASKKACDQLSLGVAAIFGPSHSSSANAVQS
ICNALGVPHIQTRWKHQVSDNKDSFYVSLYPDFSSLSRAILDLVQFFKWKTVTVVYDDST
GLIRLQELIKAPSRYNLRLKIRQLPADTKDAKPLLKEMKRGKEFHVIFDCSHEMAAGILK
QALAMGMMTEYYHYIFTTLDLFALDVEPYRYSGVNMTGFRILNTENTQVSSIIEKWSMER
LQAPPKPDSGLLDGFMTTDAALMYDAVHVVSVAVQQFPQMTVSSLQCNRHKPWRFGTRFM
SLIKEAHWEGLTGRITFNKTNGLRTDFDLDVISLKEEGLEKIGTWDPASGLNMTESQKGK
PANITDSLSNRSLIVTTILEEPYVLFKKSDKPLYGNDRFEGYCIDLLRELSTILGFTYEI
RLVEDGKYGAQDDANGQWNGMVRELIDHKADLAVAPLAITYVREKVIDFSKPFMTLGISI
LYRKPNGTNPGVFSFLNPLSPDIWMYILLAYLGVSCVLFVIARF
(서열 번호: 104)
GluK2 폴리펩티드는 서열 번호: 105의 아미노산 서열을 가질 수 있거나 또는 서열 번호: 105의 아미노산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있는데, 이것은 아래에 도시된다 (UniProt Q13002-4; 글루타민산염 수용체 이온성, 카이네이트 2의 GRIK2_HUMAN 동종형 4):
MKIIFPILSNPVFRRTVKLLLCLLWIGYSQGTTHVLRFGGIFEYVESGPMGAEELAFRFA
VNTINRNRTLLPNTTLTYDTQKINLYDSFEASKKACDQLSLGVAAIFGPSHSSSANAVQS
ICNALGVPHIQTRWKHQVSDNKDSFYVSLYPDFSSLSRAILDLVQFFKWKTVTVVYDDST
GLIRLQELIKAPSRYNLRLKIRQLPADTKDAKPLLKEMKRGKEFHVIFDCSHEMAAGILK
QALAMGMMTEYYHYIFTTLDLFALDVEPYRYSGVNMTGFRILNTENTQVSSIIEKWSMER
LQAPPKPDSGLLDGFMTTDAALMYDAVHVVSVAVQQFPQMTVSSLQCNRHKPWRFGTRFM
SLIKEAHWEGLTGRITFNKTNGLRTDFDLDVISLKEEGLEKIGTWDPASGLNMTESQKGK
PANITDSLSNRSLIVTTILEEPYVLFKKSDKPLYGNDRFEGYCIDLLRELSTILGFTYEI
RLVEDGKYGAQDDANGQWNGMVRELIDHKADLAVAPLAITYVREKVIDFSKPFMTLGISI
LYRKPNGSELMPKALSTRIVGGIWWFFTLIIISSYTANLAAFLTVERMESPIDSADDLAK
QTKIEYGAVEDGATMTFFKKSKISTYDKMWAFMSSRRQSVLVKSNEEGIQRVLTSDYAFL
MESTTIEFVTQRNCNLTQIGGLIDSKGYGVGTPMGSPYRDKITIAILQLQEEGKLHMMKE
KWWRGNGCPEEESKEASALGVQNIGGIFIVLAAGLVLSVFVAVGEFLYKSKKNAQLEKRS
FCSAMVEELRMSLKCQRRLKHKPQAPVIVKTEEVINMHTFNDRRLPGKETMA
(서열 번호: 105)
GluK2 폴리펩티드는 서열 번호: 106의 아미노산 서열을 가질 수 있거나 또는 서열 번호: 106의 아미노산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있는데, 이것은 아래에 도시된다 (UniProt Q13002-5; 글루타민산염 수용체 이온성, 카이네이트 2의 GRIK2_HUMAN 동종형 5):
MKIIFPILSNPVFRRTVKLLLCLLWIGYSQGTTHVLRFGGIFEYVESGPMGAEELAFRFA
VNTINRNRTLLPNTTLTYDTQKINLYDSFEASKKACDQLSLGVAAIFGPSHSSSANAVQS
ICNALGVPHIQTRWKHQVSDNKDSFYVSLYPDFSSLSRAILDLVQFFKWKTVTVVYDDST
GLIRLQELIKAPSRYNLRLKIRQLPADTKDAKPLLKEMKRGKEFHVIFDCSHEMAAGILK
QALAMGMMTEYYHYIFTTLDLFALDVEPYRYSGVNMTGFRILNTENTQVSSIIEKWSMER
LQAPPKPDSGLLDGFMTTDAALMYDAVHVVSVAVQQFPQMTVSSLQCNRHKPWRFGTRFM
SLIKEAHWEGLTGRITFNKTNGLRTDFDLDVISLKEEGLEKIGTWDPASGLNMTESQKGK
PANITDSLSNRSLIVTTILEEPYVLFKKSDKPLYGNDRFEGYCIDLLRELSTILGFTYEI
RLVEDGKYGAQDDANGQWNGMVRELIDHKADLAVAPLAITYVREKVIDFSKPFMTLGISI
LYRKPNGTNPGVFSFLNPLSPDIWMYILLAYLGVSCVLFVIARFSPYEWYNPHPCNPDSD
VVENNFTLLNSFWFGVGALMQQGSELMPKALSTRIVGGIWWFFTLIIISSYTANLAAFLT
VERMESPIDSADDLAKQTKIEYGAVEDGATMTFFKKSKISTYDKMWAFMSSRRQSVLVKS
NEEGIQRVLTSDYAFLMESTTIEFVTQRNCNLTQIGGLIDSKGYGVGTPMGSPYRDKITI
AILQLQEEGKLHMMKEKWWRGNGCPEEESKEASALGVQNIGGIFIVLAAGLVLSVFVAVG
EFLYKSKKNAQLEKRAKTKLPQDYVFLPILESVSISTVLSSSPSSSSLSSCS
(서열 번호: 106)
GluK2 폴리펩티드는 서열 번호: 107의 아미노산 서열을 가질 수 있거나 또는 서열 번호: 107의 아미노산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있는데, 이것은 아래에 도시된다 (UniProt Q13002-6; 글루타민산염 수용체 이온성, 카이네이트 2의 GRIK2_HUMAN 동종형 6):
MKIIFPILSNPVFRRTVKLLLCLLWIGYSQGTTHVLRFGGIFEYVESGPMGAEELAFRFA
VNTINRNRTLLPNTTLTYDTQKINLYDSFEASKKACDQLSLGVAAIFGPSHSSSANAVQS
ICNALGVPHIQTRWKHQVSDNKDSFYVSLYPDFSSLSRAILDLVQFFKWKTVTVVYDDST
GLIRLQELIKAPSRYNLRLKIRQLPADTKDAKPLLKEMKRGKEFHVIFDCSHEMAAGILK
QALAMGMMTEYYHYIFTTLDLFALDVEPYRYSGVNMTGFRILNTENTQVSSIIEKWSMER
LQAPPKPDSGLLDGFMTTDAALMYDAVHVVSVAVQQFPQMTVSSLQCNRHKPWRFGTRFM
SLIKEAHWEGLTGRITFNKTNGLRTDFDLDVISLKEEGLEKIGTWDPASGLNMTESQKGK
PANITDSLSNRSLIVTTILEEPYVLFKKSDKPLYGNDRFEGYCIDLLRELSTILGFTYEI
RLVEDGKYGAQDDANGQWNGMVRELIDHKSKISTYDKMWAFMSSRRQSVLVKSNEEGIQR
VLTSDYAFLMESTTIEFVTQRNCNLTQIGGLIDSKGYGVGTPMGSPYRDKITIAILQLQE
EGKLHMMKEKWWRGNGCPEEESKEASALGVQNIGGIFIVLAAGLVLSVFVAVGEFLYKSK
KNAQLEKESSIWLVPPYHPDTV
(서열 번호: 107)
GluK2 폴리펩티드는 서열 번호: 108의 아미노산 서열을 가질 수 있거나 또는 서열 번호: 108의 아미노산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있는데, 이것은 아래에 도시된다 (UniProt Q13002-7; 글루타민산염 수용체 이온성, 카이네이트 2의 GRIK2_HUMAN 동종형 7):
MKIIFPILSNPVFRRTVKLLLCLLWIGYSQGTTHVLRFGGIFEYVESGPMGAEELAFRFA
VNTINRNRTLLPNTTLTYDTQKINLYDSFEASKKACDQLSLGVAAIFGPSHSSSANAVQS
ICNALGVPHIQTRWKHQVSDNKDSFYVSLYPDFSSLSRAILDLVQFFKWKTVTVVYDDST
GLIRLQELIKAPSRYNLRLKIRQLPADTKDAKPLLKEMKRGKEFHVIFDCSHEMAAGILK
QALAMGMMTEYYHYIFTTLDLFALDVEPYRYSGVNMTGFRILNTENTQVSSIIEKWSMER
LQAPPKPDSGLLDGFMTTDAALMYDAVHVVSVAVQQFPQMTVSSLQCNRHKPWRFGTRFM
SLIKEAHWEGLTGRITFNKTNGLRTDFDLDVISLKEEGLEKIGTWDPASGLNMTESQKGK
PANITDSLSNRSLIVTTILEEPYVLFKKSDKPLYGNDRFEGYCIDLLRELSTILGFTYEI
RLVEDGKYGAQDDANGQWNGMVRELIDHKSVLVKSNEEGIQRVLTSDYAFLMESTTIEFV
TQRNCNLTQIGGLIDSKGYGVGTPMGSPYRDKITIAILQLQEEGKLHMMKEKWWRGNGCP
EEESKEASALGVQNIGGIFIVLAAGLVLSVFVAVGEFLYKSKKNAQLEKRAKTKLPQDYV
FLPILESVSISTVLSSSPSSSSLSSCS
(서열 번호: 108)
GluK2 폴리펩티드는 서열 번호: 109의 아미노산 서열을 가질 수 있거나 또는 서열 번호: 109의 아미노산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있는데, 이것은 아래에 도시된다 (NP_001104738.2; 글루타민산염 수용체 이온성, 카이네이트 2의 GRIK2_MOUSE 동종형 1 전구체):
MKIISPVLSNLVFSRSIKVLLCLLWIGYSQGTTHVLRFGGIFEYVESGPMGAEELAFRFAVNTINRNRTLLPNTTLTYDTQKINLYDSFEASKKACDQLSLGVAAIFGPSHSSSANAVQSICNALGVPHIQTRWKHQVSDNKDSFYVSLYPDFSSLSRAILDLVQFFKWKTVTVVYDDSTGLIRLQELIKAPSRYNLRLKIRQLPADTKDAKPLLKEMKRGKEFHVIFDCSHEMAAGILKQALAMGMMTEYYHYIFTTLDLFALDVEPYRYSGVNMTGFRILNTENTQVSSIIEKWSMERLQAPPKPDSGLLDGFMTTDAALMYDAVHVVSVAVQQFPQMTVSSLQCNRHKPWRFGTRFMSLIKEAHWEGLTGRITFNKTNGLRTDFDLDVISLKEEGLEKIGTWDPSSGLNMTESQKGKPANITDSLSNRSLIVTTILEEPYVLFKKSDKPLYGNDRFEGYCIDLLRELSTILGFTYEIRLVEDGKYGAQDDVNGQWNGMVRELIDHKADLAVAPLAITYVREKVIDFSKPFMTLGISILYRKPNGTNPGVFSFLNPLSPDIWMYILLAYLGVSCVLFVIARFSPYEWYNPHPCNPDSDVVENNFTLLNSFWFGVGALMQQGSELMPKALSTRIVGGIWWFFTLIIISSYTANLAAFLTVERMESPIDSADDLAKQTKIEYGAVEDGATMTFFKKSKISTYDKMWAFMSSRRQSVLVKSNEEGIQRVLTSDYAFLMESTTIEFVTQRNCNLTQIGGLIDSKGYGVGTPMGSPYRDKITIAILQLQEEGKLHMMKEKWWRGNGCPEEESKEASALGVQNIGGIFIVLAAGLVLSVFVAVGEFLYKSKKNAQLEKRSFCSAMVEELRMSLKCQRRLKHKPQAPVIVKTEEVINMHTFNDRRLPGKETMA
(서열 번호: 109)
GluK2 폴리펩티드는 서열 번호: 110의 아미노산 서열을 가질 수 있거나 또는 서열 번호: 110의 아미노산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있는데, 이것은 아래에 도시된다 (NP_034479.3; 글루타민산염 수용체 이온성, 카이네이트 2의 GRIK2_MOUSE 동종형 2 전구체):
MKIISPVLSNLVFSRSIKVLLCLLWIGYSQGTTHVLRFGGIFEYVESGPMGAEELAFRFAVNTINRNRTL
LPNTTLTYDTQKINLYDSFEASKKACDQLSLGVAAIFGPSHSSSANAVQSICNALGVPHIQTRWKHQVSDNKDSFYVSLYPDFSSLSRAILDLVQFFKWKTVTVVYDDSTGLIRLQELIKAPSRYNLRLKIRQLPADTKDAKPLLKEMKRGKEFHVIFDCSHEMAAGILKQALAMGMMTEYYHYIFTTLDLFALDVEPYRYSGVNMTGFRILNTENTQVSSIIEKWSMERLQAPPKPDSGLLDGFMTTDAALMYDAVHVVSVAVQQFPQMTVSSLQCNRHKPWRFGTRFMSLIKEAHWEGLTGRITFNKTNGLRTDFDLDVISLKEEGLEKIGTWDPSSGLNMTESQKGKPANITDSLSNRSLIVTTILEEPYVLFKKSDKPLYGNDRFEGYCIDLLRELSTILGFTYEIRLVEDGKYGAQDDVNGQWNGMVRELIDHKADLAVAPLAITYVREKVIDFSKPFMTLGISILYRKPNGTNPGVFSFLNPLSPDIWMYILLAYLGVSCVLFVIARFSPYEWYNPHPCNPDSDVVENNFTLLNSFWFGVGALMQQGSELMPKALSTRIVGGIWWFFTLIIISSYTANLAAFLTVERMESPIDSADDLAKQTKIEYGAVEDGATMTFFKKSKISTYDKMWAFMSSRRQSVLVKSNEEGIQRVLTSDYAFLMESTTIEFVTQRNCNLTQIGGLIDSKGYGVGTPMGSPYRDKITIAILQLQEEGKLHMMKEKWWRGNGCPEEESKEASALGVQNIGGIFIVLAAGLVLSVFVAVGEFLYKSKKNAQLEKESSIWLVPPYHPDTV
(서열 번호: 110)
GluK2 폴리펩티드는 서열 번호: 111의 아미노산 서열을 가질 수 있거나 또는 서열 번호: 111의 아미노산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있는데, 이것은 아래에 도시된다 (NP_001345795.2; 글루타민산염 수용체 이온성, 카이네이트 2의 GRIK2_MOUSE 동종형 1 전구체):
MKIISPVLSNLVFSRSIKVLLCLLWIGYSQGTTHVLRFGGIFEYVESGPMGAEELAFRFAVNTINRNRTL
LPNTTLTYDTQKINLYDSFEASKKACDQLSLGVAAIFGPSHSSSANAVQSICNALGVPHIQTRWKHQVSDNKDSFYVSLYPDFSSLSRAILDLVQFFKWKTVTVVYDDSTGLIRLQELIKAPSRYNLRLKIRQLPADTKDAKPLLKEMKRGKEFHVIFDCSHEMAAGILKQALAMGMMTEYYHYIFTTLDLFALDVEPYRYSGVNMTGFRILNTENTQVSSIIEKWSMERLQAPPKPDSGLLDGFMTTDAALMYDAVHVVSVAVQQFPQMTVSSLQCNRHKPWRFGTRFMSLIKEAHWEGLTGRITFNKTNGLRTDFDLDVISLKEEGLEKIGTWDPSSGLNMTESQKGKPANITDSLSNRSLIVTTILEEPYVLFKKSDKPLYGNDRFEGYCIDLLRELSTILGFTYEIRLVEDGKYGAQDDVNGQWNGMVRELIDHKADLAVAPLAITYVREKVIDFSKPFMTLGISILYRKPNGTNPGVFSFLNPLSPDIWMYILLAYLGVSCVLFVIARFSPYEWYNPHPCNPDSDVVENNFTLLNSFWFGVGALMQQGSELMPKALSTRIVGGIWWFFTLIIISSYTANLAAFLTVERMESPIDSADDLAKQTKIEYGAVEDGATMTFFKKSKISTYDKMWAFMSSRRQSVLVKSNEEGIQRVLTSDYAFLMESTTIEFVTQRNCNLTQIGGLIDSKGYGVGTPMGSPYRDKITIAILQLQEEGKLHMMKEKWWRGNGCPEEESKEASALGVQNIGGIFIVLAAGLVLSVFVAVGEFLYKSKKNAQLEKRSFCSAMVEELRMSLKCQRRLKHKPQAPVIVKTEEVINMHTFNDRRLPGKETMA
(서열 번호: 111)
GluK2 폴리펩티드는 서열 번호: 112의 아미노산 서열을 가질 수 있거나 또는 서열 번호: 112의 아미노산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있는데, 이것은 아래에 도시된다 (XP_014992481.1; GRIK2_RHESUS MACAQUE 동종형 X1, 글루타민산염 수용체 이온성, 카이네이트 2):
MKIIFPILSNPVFRRTVKLLLCLLWIGYSQGTTHVLRFGGIFEYVESGPMGAEELAFRFAVNTINRNRTL
LPNTTLTYDTQKINLYDSFEASKKACDQLSLGVAAIFGPSHSSSANAVQSICNALGVPHIQTRWKHQVSDNKDSFYVSLYPDFSSLSRAILDLVQFFKWKTVTVVYDDSTGLIRLQELIKAPSRYNLRLKIRQLPADTKDAKPLLKEMKRGKEFHVIFDCSHEMAAGILKQALAMGMMTEYYHYIFTTLDLFALDVEPYRYSGVNMTGFRILNTENTQVSSIIEKWSMERLQAPPKPDSGLLDGFMTTDAALMYDAVHVVSVAVQQFPQMTVSSLQCNRHKPWRFGTRFMSLIKEAHWEGLTGRITFNKTNGLRTDFDLDVISLKEEGLEKIGTWDPASGLNMTESQKGKPANITDSLSNRSLIVTTILEEPYVLFKKSDKPLYGNDRFEGYCIDLLRELSTILGFTYEIRLVEDGKYGAQDDANGQWNGMVRELIDHKADLAVAPLAITYVREKVIDFSKPFMTLGISILYRKPNGTNPGVFSFLNPLSPDIWMYILLAYLGVSCVLFVIARFSPYEWYNPHPCNPDSDVVENNFTLLNSFWFGVGALMQQGSELMPKALSTRIVGGIWWFFTLIIISSYTANLAAFLTVERMESPIDSADDLAKQTKIEYGAVEDGATMTFFKKSKISTYDKMWAFMSSRRQSVLVKSNEEGIQRVLTSDYAFLMESTTIEFVTQRNCNLTQIGGLIDSKGYGVGTPMGSPYRDKITIAILQLQEEGKLHMMKEKWWRGNGCPEEESKEASALGVQNIGGIFIVLAAGLVLSVFVAVGEFLYKSKKNAQLEKRSFCSAMVEELRMSLKCQRRLKHKPQAPVIVKTEEVINMHTFNDRRLPGKETMA
(서열 번호: 112)
GluK2 폴리펩티드는 서열 번호: 113의 아미노산 서열을 가질 수 있거나 또는 서열 번호: 113의 아미노산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있는데, 이것은 아래에 도시된다 (XP_014992483.1; GRIK2_RHESUS MACAQUE 동종형 X1, 글루타민산염 수용체 이온성, 카이네이트 2):
MKIIFPILSNPVFRRTVKLLLCLLWIGYSQGTTHVLRFGGIFEYVESGPMGAEELAFRFAVNTINRNRTL
LPNTTLTYDTQKINLYDSFEASKKACDQLSLGVAAIFGPSHSSSANAVQSICNALGVPHIQTRWKHQVSDNKDSFYVSLYPDFSSLSRAILDLVQFFKWKTVTVVYDDSTGLIRLQELIKAPSRYNLRLKIRQLPADTKDAKPLLKEMKRGKEFHVIFDCSHEMAAGILKQALAMGMMTEYYHYIFTTLDLFALDVEPYRYSGVNMTGFRILNTENTQVSSIIEKWSMERLQAPPKPDSGLLDGFMTTDAALMYDAVHVVSVAVQQFPQMTVSSLQCNRHKPWRFGTRFMSLIKEAHWEGLTGRITFNKTNGLRTDFDLDVISLKEEGLEKIGTWDPASGLNMTESQKGKPANITDSLSNRSLIVTTILEEPYVLFKKSDKPLYGNDRFEGYCIDLLRELSTILGFTYEIRLVEDGKYGAQDDANGQWNGMVRELIDHKADLAVAPLAITYVREKVIDFSKPFMTLGISILYRKPNGTNPGVFSFLNPLSPDIWMYILLAYLGVSCVLFVIARFSPYEWYNPHPCNPDSDVVENNFTLLNSFWFGVGALMQQGSELMPKALSTRIVGGIWWFFTLIIISSYTANLAAFLTVERMESPIDSADDLAKQTKIEYGAVEDGATMTFFKKSKISTYDKMWAFMSSRRQSVLVKSNEEGIQRVLTSDYAFLMESTTIEFVTQRNCNLTQIGGLIDSKGYGVGTPMGSPYRDKITIAILQLQEEGKLHMMKEKWWRGNGCPEEESKEASALGVQNIGGIFIVLAAGLVLSVFVAVGEFLYKSKKNAQLEKRSFCSAMVEELRMSLKCQRRLKHKPQAPVIVKTEEVINMHTFNDRRLPGKETMA
(서열 번호: 113)
GluK2 폴리펩티드는 서열 번호: 114의 아미노산 서열을 가질 수 있거나 또는 서열 번호: 114의 아미노산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있는데, 이것은 아래에 도시된다 (NP_062182.1; 글루타민산염 수용체 이온성, 카이네이트 2의 GRIK2_RAT 전구체):
MKIISPVLSNLVFSRSIKVLLCLLWIGYSQGTTHVLRFGGIFEYVESGPMGAEELAFRFAVNTINRNRTL
LPNTTLTYDTQKINLYDSFEASKKACDQLSLGVAAIFGPSHSSSANAVQSICNALGVPHIQTRWKHQVSDNKDSFYVSLYPDFSSLSRAILDLVQFFKWKTVTVVYDDSTGLIRLQELIKAPSRYNLRLKIRQLPADTKDAKPLLKEMKRGKEFHVIFDCSHEMAAGILKQALAMGMMTEYYHYIFTTLDLFALDVEPYRYSGVNMTGFRILNTENTQVSSIIEKWSMERLQAPPKPDSGLLDGFMTTDAALMYDAVHVVSVAVQQFPQMTVSSLQCNRHKPWRFGTRFMSLIKEAHWEGLTGRITFNKTNGLRTDFDLDVISLKEEGLEKIGTWDPASGLNMTESQKGKPANITDSLSNRSLIVTTILEEPYVLFKKSDKPLYGNDRFEGYCIDLLRELSTILGFTYEIRLVEDGKYGAQDDVNGQWNGMVRELIDHKADLAVAPLAITYVREKVIDFSKPFMTLGISILYRKPNGTNPGVFSFLNPLSPDIWMYVLLACLGVSCVLFVIARFSPYEWYNPHPCNPDSDVVENNFTLLNSFWFGVGALMRQGSELMPKALSTRIVGGIWWFFTLIIISSYTANLAAFLTVERMESPIDSADDLAKQTKIEYGAVEDGATMTFFKKSKISTYDKMWAFMSSRRQSVLVKSNEEGIQRVLTSDYAFLMESTTIEFVTQRNCNLTQIGGLIDSKGYGVGTPMGSPYRDKITIAILQLQEEGKLHMMKEKWWRGNGCPEEESKEASALGVQNIGGIFIVLAAGLVLSVFVAVGEFLYKSKKNAQLEKRSFCSAMVEELRMSLKCQRRLKHKPQAPVIVKTEEVINMHTFNDRRLPGKETMA
(서열 번호: 114)
Grik2 mRNA는 5'와 3' 비번역 영역 (UTR)을 내포하고 서열 번호: 115의 핵산 서열을 갖는 폴리뉴클레오티드일 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 115의 핵산 서열 (RefSeq NM_021956.1:4592 호모 사피엔스 (Homo sapiens) 글루타민산염 이온성 수용체 카이네이트 유형 아단위 2 (GRIK2), 전사체 변이체 1, mRNA)에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
Grik2 mRNA는 서열 번호: 116의 핵산 서열을 갖는 폴리뉴클레오티드일 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 116의 핵산 서열 (RefSeq NM_021956.4:294-3020 호모 사피엔스 (Homo sapiens) 글루타민산염 이온성 수용체 카이네이트 유형 아단위 2 (GRIK2), 전사체 변이체 1, mRNA)에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
부가적으로 또는 대안으로, Grik2 mRNA는 서열 번호: 117의 핵산 서열을 갖는 폴리뉴클레오티드일 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 117의 핵산 서열 (RefSeq NM_175768.3:294-2903 호모 사피엔스 (Homo sapiens) 글루타민산염 이온성 수용체 카이네이트 유형 아단위 2 (GRIK2), 전사체 변이체 2, mRNA)에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
부가적으로 또는 대안으로, Grik2 mRNA는 서열 번호: 118의 핵산 서열을 갖는 폴리뉴클레오티드일 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 118의 핵산 서열 (RefSeq NM_001166247.1:294-2972 호모 사피엔스 (Homo sapiens) 글루타민산염 이온성 수용체 카이네이트 유형 아단위 2 (GRIK2), 전사체 변이체 3, mRNA)에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
부가적으로 또는 대안으로, Grik2 mRNA는 서열 번호: 119의 핵산 서열을 갖는 폴리뉴클레오티드일 수 있거나, 또는 아래의 표에서 도시된 바와 같은 서열 번호: 119의 핵산 서열 (RefSeq NM_001111268.2 무스 무스쿨루스 (Mus musculus) 글루타민산염 이온성 수용체 카이네이트 유형 아단위 2 (GRIK2), 전사체 변이체 4, mRNA)에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
부가적으로 또는 대안으로, Grik2 mRNA는 서열 번호: 120의 핵산 서열을 갖는 폴리뉴클레오티드일 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 120의 핵산 서열 (RefSeq NM_010349.4 무스 무스쿨루스 (Mus musculus) 글루타민산염 이온성 수용체 카이네이트 유형 아단위 2 (GRIK2), 전사체 변이체 5, mRNA)에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
부가적으로 또는 대안으로, Grik2 mRNA는 서열 번호: 121의 핵산 서열을 갖는 폴리뉴클레오티드일 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 121의 핵산 서열 (RefSeq NM_ 001358866 무스 무스쿨루스 (Mus musculus) 글루타민산염 이온성 수용체 카이네이트 유형 아단위 2 (GRIK2), 전사체 변이체 6, mRNA)에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
부가적으로 또는 대안으로, Grik2 mRNA는 서열 번호: 122의 핵산 서열을 갖는 폴리뉴클레오티드일 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 122의 핵산 서열 (RefSeq XM_015136995.2 마카카 물라타 (Macaca mulatta) 글루타민산염 이온성 수용체 카이네이트 유형 아단위 2 (GRIK2), 전사체 변이체 7, mRNA)에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
부가적으로 또는 대안으로, Grik2 mRNA는 서열 번호: 123의 핵산 서열을 갖는 폴리뉴클레오티드일 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 123의 핵산 서열 (RefSeq XM_015136997.2 마카카 물라타 (Macaca mulatta) 글루타민산염 이온성 수용체 카이네이트 유형 아단위 2 (GRIK2), 전사체 변이체 X1, mRNA)에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
부가적으로 또는 대안으로, Grik2 mRNA는 서열 번호: 124의 핵산 서열을 갖는 폴리뉴클레오티드일 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 124의 핵산 서열 (RefSeq NM_019309.2 라투스 노르베기쿠스 (Rattus norvegicus) 글루타민산염 이온성 수용체 카이네이트 유형 아단위 2 (GRIK2), mRNA)에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
부가적으로 또는 대안으로, Grik2 mRNA는 성숙 GluK2 펩티드 코딩 서열에 상응하고, 그리고 서열 번호: 125의 핵산 서열 또는 표 4에서 도시된 바와 같은, 서열 번호: 125의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 폴리뉴클레오티드를 포함한다.
개시된 방법과 조성물에 따라서, Grik2 mRNA는 5' UTR, 예컨대 예를 들면, 서열 번호: 126의 핵산 서열 또는 표 4에서 도시된 바와 같은, 서열 번호: 126의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 폴리뉴클레오티드에 의해 인코딩된 5' UTR을 포함할 수 있다.
Grik2 mRNA는 3' UTR, 예컨대 예를 들면, 서열 번호: 127의 핵산 서열 또는 표 4에서 도시된 바와 같은, 서열 번호: 127의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 폴리뉴클레오티드에 의해 인코딩된 3' UTR을 포함할 수 있다.
추가적으로, Grik2 mRNA는 Grik2 신호 펩티드 서열, 예컨대 예를 들면, 서열 번호: 128의 핵산 서열 또는 표 4에서 도시된 바와 같은, 서열 번호: 128의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에 의해 인코딩된 신호 펩티드 서열을 인코딩하는 폴리뉴클레오티드를 포함할 수 있다.
Grik2 mRNA 표적 서열
본원 발명의 ASO 작용제는 Grik2 mRNA의 하나 이상의 영역 (예를 들면, 본원에서 확인된 하나 이상의 영역), 예컨대 예를 들면, Grik2 mRNA의 번역 개시 부위 (AUG 코돈), 코딩 영역에서 서열 (예를 들면 본원에서 설명되는 엑손 1-16 중에서 하나 이상), 또는 5' UTR 또는 3' UTR을 갖는 영역을 표적으로 할 수 있다 (예를 들면, 이것에 특이적으로 혼성화할 수 있다). 이들 영역을 표적으로 함으로써, 본원 발명의 ASO 작용제는 상기 mRNA의 단백질 번역을 위한 부위로의 전위 (예를 들면, 핵으로부터 세포질로의 전위), 상기 mRNA의 GluK2 단백질로의 번역, 상기 mRNA의 스플라이싱 또는 성숙 및/또는 상기 RNA가 관여할 수 있는 독립된 촉매 활성을 포함하지만 이들에 한정되지 않는, 상기 mRNA의 정상적인 생물학적 처리를 간섭할 수 있다. 이러한 RNA 기능을 이용한 이런 간섭의 전반적인 효과는 Gluk2 단백질 발현의 간섭을 야기하고, 그것에 의하여 세포 (예를 들면, 뉴런 또는 성상 세포)에서 GluK2 발현을 감소시키거나 또는 제거하는 것이다.
Grik2 표적 서열은 안티센스 RNA에 의한 저해 또는 녹다운에 순응하는 Grik2 mRNA 서열 (예를 들면, 센스 표적 서열)의 일부 또는 영역이다. 핵산의 여러 표적 부위가, 표적화된 Grik2 전사체의 인식 부위로서 확인되었다. 아래의 표 4에서 도시된 바와 같이, Grik2 표적 부위에 혼성화하는 (또는 결합하는) 다양한 안티센스 RNA가 본 발명자들에 의해 확인되었다. Grik2 mRNA 표적 핵산은 일차 전사체 (RNA) 또는 이것을 인코딩하는 cDNA의 영역 내에 뉴클레오티드 서열을 포함한다. 당업자는 cDNA 서열이 우리딘의 티미딘으로의 치환을 제외하고, mRNA 서열과 동등하고, 그리고 본원에서 동일한 목적, 다시 말하면, Grik2 mRNA의 발현을 저해하기 위한 안티센스 올리고뉴클레오티드의 생성에 이용될 수 있다는 것을 이해할 것이다.
본원에서 개시된 방법과 조성물과 함께 이용될 수 있는 저해성 RNA 작제물 (예를 들면, 본원에서 개시된 ASO 작용제)는 예컨대 예를 들면, 서열 번호: 115-125의 Grik2 mRNA 전사체, 5' UTR (서열 번호: 126), 3' UTR (서열 번호: 127), Grik2 신호 펩티드를 인코딩하는 핵산 서열 (서열 번호: 128), Grik2 mRNA의 엑손 1 (서열 번호: 129), Grik2 mRNA의 엑손 2 (서열 번호: 130), Grik2 mRNA의 엑손 3 (서열 번호: 131), Grik2 mRNA의 엑손 4 (서열 번호: 132), Grik2 mRNA의 엑손 5 (서열 번호: 133), Grik2 mRNA의 엑손 6 (서열 번호: 134), Grik2 mRNA의 엑손 7 (서열 번호: 135), Grik2 mRNA의 엑손 8 (서열 번호: 136), Grik2 mRNA의 엑손 9 (서열 번호: 137), Grik2 mRNA의 엑손 10 (서열 번호: 138), Grik2 mRNA의 엑손 11 (서열 번호: 139), Grik2 mRNA의 엑손 12 (서열 번호: 140), Grik2 mRNA의 엑손 13 (서열 번호: 141), Grik2 mRNA의 엑손 14 (서열 번호: 142), Grik2 mRNA의 엑손 15 (서열 번호: 143) 및 Grik2 mRNA의 엑손 16 (서열 번호: 144) 중에서 하나의 적어도 일부 내에, Grik2 mRNA의 하나 이상 (예를 들면, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 12, 13, 14, 15, 16, 17, 18, 19개, 또는 그 초과)의 표적 영역에 결합할 수 있는 (예를 들면, 상보성 염기 대합에 의해) ASO 작용제를 포함한다. 서열 번호: 115 또는 서열 번호: 116의 적어도 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 표 2 또는 표 3에서 열거된 ASO 작용제에서 선택될 수 있다.
예를 들면, 본원 발명의 재조합 ASO 작용제는 서열 번호: 115의 적어도 일부 또는 영역 내에 뉴클레오티드 서열에 상보적인 뉴클레오티드 서열을 포함한다. 다른 실례에서, ASO 작용제는 서열 번호: 116의 적어도 일부 또는 영역 내에 뉴클레오티드 서열에 상보적인 뉴클레오티드 서열을 포함한다.
추가 실례에서, Grik2 mRNA를 표적으로 하는 본원 발명의 ASO 작용제는 5' UTR (서열 번호: 126)의 적어도 일부 또는 영역 내에 뉴클레오티드 서열에 상보적인 뉴클레오티드 서열을 포함한다. 다른 실례에서, Grik2 mRNA를 표적으로 하는 본원 발명의 ASO 작용제는 3' UTR (서열 번호: 127)의 적어도 일부 또는 영역 내에 뉴클레오티드 서열에 상보적인 뉴클레오티드 서열을 포함한다.
개시된 ASO 작용제는 Grik2 mRNA의 하나 이상의 엑손, 예컨대 예를 들면, 서열 번호: 115의 핵산 서열 또는 서열 번호: 115의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 Grik2 mRNA의 하나 이상의 엑손에 혼성화할 수 있다. 따라서, ASO 작용제는 Grik2 mRNA의 엑손 1, 예컨대 예를 들면, 서열 번호: 115의 뉴클레오티드 위치 1-408에 위치된 Grik2 mRNA의 엑손 1의 적어도 일부 또는 영역 내에 혼성화할 수 있다. Grik2 mRNA의 엑손 1의 서열은 서열 번호: 129의 핵산 서열 또는 표 4에서 도시된 바와 같은, 서열 번호: 129의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
예를 들면, 본원에서 개시된 ASO 작용제를 이용한 표적화에 대해 예기된 Grik2 표적 핵산은 뉴클레오티드 197-217 (서열 번호: 115), 215-235 (서열 번호: 115), 232-251 (서열 번호: 115), 232-252 (서열 번호: 115), 227-247 (서열 번호: 115), 29-48 (서열 번호: 116), 322-341 (서열 번호: 115), 29-49 (서열 번호: 116), 322-342 (서열 번호: 115), 182-202 (서열 번호: 115), 226-246 (서열 번호: 115), 253-272 (서열 번호: 115), 253-273 (서열 번호: 115), 139-159 (서열 번호: 115), 176-196 (서열 번호: 115), 241-261 (서열 번호: 115), 195-215 (서열 번호: 115), 42-62 (서열 번호: 115), 196-216 (서열 번호: 115), 또는 30-49 (서열 번호: 115)를 포함한다. 추가적으로, Grik2 ASO 작용제는 뉴클레오티드 197-217 (서열 번호: 115), 215-235 (서열 번호: 115), 232-251 (서열 번호: 115), 232-252 (서열 번호: 115), 227-247 (서열 번호: 115), 29-48 (서열 번호: 116), 322-341 (서열 번호: 115), 29-49 (서열 번호: 116), 322-342 (서열 번호: 115), 182-202 (서열 번호: 115), 226-246 (서열 번호: 115), 253-272 (서열 번호: 115), 253-273 (서열 번호: 115), 139-159 (서열 번호: 115), 176-196 (서열 번호: 115), 241-261 (서열 번호: 115), 195-215 (서열 번호: 115), 42-62 (서열 번호: 115), 196-216 (서열 번호: 115), 30-49 (서열 번호: 115), 또는 이들의 단편 또는 일부 내에 Grik2 mRNA에 혼성화할 수 있다.
서열 번호: 116 또는 서열 번호: 115의 엑손 1의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO 작용제는 siRNA TJ (서열 번호: 21), siRNA TG (서열 번호: 23), siRNA TF (서열 번호: 24), siRNA TE (서열 번호: 25), siRNA TD (서열 번호: 26), siRNA TC (서열 번호: 28), siRNA CK (서열 번호: 29), siRNA CX (서열 번호: 42), siRNA CY (서열 번호: 43), siRNA D0 (서열 번호: 45), siRNA D1 (서열 번호: 46), siRNA D3 (서열 번호: 48), siRNA XZ (서열 번호: 54), siRNA Y0 (서열 번호: 55), siRNA GF (서열 번호: 64), siRNA ZZ (서열 번호: 100), siRNA GE (서열 번호: 65), siRNA GH (서열 번호: 66), 또는 siRNA YB (서열 번호: 67), 또는 siRNA TJ (서열 번호: 21), siRNA TG (서열 번호: 23), siRNA TF (서열 번호: 24), siRNA TE (서열 번호: 25), siRNA TD (서열 번호: 26), siRNA TC (서열 번호: 28), siRNA CK (서열 번호: 29), siRNA CX (서열 번호: 42), siRNA CY (서열 번호: 43), siRNA D0 (서열 번호: 45), siRNA D1 (서열 번호: 46), siRNA D3 (서열 번호: 48), siRNA XZ (서열 번호: 54), siRNA Y0 (서열 번호: 55), siRNA GF (서열 번호: 64), siRNA ZZ (서열 번호: 100), siRNA GE (서열 번호: 65), siRNA GH (서열 번호: 66), 또는 siRNA YB (서열 번호: 67) 중에서 하나에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 안티센스 올리고뉴클레오티드에서 선택될 수 있다. 서열 번호: 116 또는 서열 번호: 115의 엑손 1의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO 작용제는 적어도 10% (예를 들면, 적어도 10%, 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 단백질 녹다운을 나타낼 수 있다. 추가적으로, Grik2 안티센스 올리고뉴클레오티드는 siRNA TJ (서열 번호: 21), siRNA TG (서열 번호: 23), siRNA TF (서열 번호: 24), siRNA TE (서열 번호: 25), siRNA TD (서열 번호: 26), siRNA TC (서열 번호: 28), siRNA CK (서열 번호: 29), siRNA CX (서열 번호: 42), siRNA CY (서열 번호: 43), siRNA D0 (서열 번호: 45), siRNA D1 (서열 번호: 46), siRNA D3 (서열 번호: 48), siRNA XZ (서열 번호: 54), siRNA Y0 (서열 번호: 55), siRNA GF (서열 번호: 64), siRNA ZZ (서열 번호: 100), siRNA GE (서열 번호: 65), siRNA GH (서열 번호: 66), 또는 siRNA YB (서열 번호: 67), 또는 여기에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 안티센스 올리고뉴클레오티드에서 선택될 수 있고, 그리고 적어도 10% (예를 들면, 적어도 10%, 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다.
추가적으로, ASO 작용제는 Grik2 mRNA의 엑손 2, 예컨대 예를 들면, 서열 번호: 115의 뉴클레오티드 위치 409-576에 위치된 Grik2 mRNA의 엑손 2의 적어도 일부 또는 영역 내에 혼성화할 수 있다. Grik2 mRNA의 엑손 2의 서열은 서열 번호: 130의 핵산 서열 또는 표 4에서 도시된 바와 같은, 서열 번호: 130의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
예를 들면, 본원에서 개시된 ASO 작용제를 이용한 표적화에 대해 예기된 Grik2 표적 핵산은 서열 번호: 115의 뉴클레오티드 501-521 또는 서열 번호: 116의 뉴클레오티드 208-228, 또는 이들의 단편 또는 일부를 포함한다.
서열 번호: 116 또는 서열 번호: 115의 엑손 2의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO 작용제는 siRNA G0 (서열 번호: 1), 또는 siRNA G0 (서열 번호: 1)에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 안티센스 올리고뉴클레오티드이다. 다른 구체예에서, 서열 번호: 116 또는 서열 번호: 115의 엑손 2의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 안티센스 올리고뉴클레오티드는 75% 초과의 GluK2 녹다운을 나타낸다. 또 다른 구체예에서, Grik2 안티센스 올리고뉴클레오티드는 siRNA G0 (서열 번호: 1), 또는 여기에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 안티센스 올리고뉴클레오티드이고, 그리고 75% 초과 (예를 들면, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 GluK2 녹다운을 나타낸다.
ASO 작용제는 또한, Grik2 mRNA의 엑손 3, 예컨대 예를 들면, 서열 번호: 115의 뉴클레오티드 위치 577-834에 위치된 Grik2 mRNA의 엑손 3의 적어도 일부 또는 영역 내에 혼성화할 수 있다. Grik2 mRNA의 엑손 3의 서열은 서열 번호: 131의 핵산 서열 또는 표 4에서 도시된 바와 같은, 서열 번호: 131의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
예를 들면, 본원에서 개시된 ASO 작용제를 이용한 표적화에 대해 예기된 Grik2 표적 핵산은 서열 번호: 116의 뉴클레오티드 307-327 또는 서열 번호: 115의 뉴클레오티드 600-620, 서열 번호: 116의 뉴클레오티드 352-372 또는 서열 번호: 115의 뉴클레오티드 645-665, 서열 번호: 116의 뉴클레오티드 381-400 또는 서열 번호: 115의 뉴클레오티드 674-693, 서열 번호: 116의 뉴클레오티드 381-401 또는 서열 번호: 115의 뉴클레오티드 674-694, 서열 번호: 116의 뉴클레오티드 380-400 또는 서열 번호: 115의 뉴클레오티드 673-693, 서열 번호: 116의 뉴클레오티드 534-554 또는 서열 번호: 115의 뉴클레오티드 827-847, 서열 번호: 116의 뉴클레오티드 308-328 또는 서열 번호: 115의 뉴클레오티드 601-621, 서열 번호: 116의 뉴클레오티드 396-416 또는 서열 번호: 115의 뉴클레오티드 689-709, 서열 번호: 116의 뉴클레오티드 355-375 또는 서열 번호: 115의 뉴클레오티드 648-668, 서열 번호: 116의 뉴클레오티드 357-377 또는 서열 번호: 115의 뉴클레오티드 650-670, 서열 번호: 116의 뉴클레오티드 424-444 또는 서열 번호: 115의 뉴클레오티드 717-737, 서열 번호: 116의 뉴클레오티드 429-449 또는 서열 번호: 115의 뉴클레오티드 722-742, 또는 이들의 단편 또는 일부를 포함한다.
서열 번호: 116 또는 서열 번호: 115의 엑손 3의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO 작용제는 siRNA TV (서열 번호: 2), siRNA TU (서열 번호: 3), siRNA CL (서열 번호: 30), siRNA CM (서열 번호: 31), siRNA CR (서열 번호: 36), siRNA CV (서열 번호: 40), siRNA Y4 (서열 번호: 59), siRNA MP (서열 번호: 76), siRNA MW (서열 번호: 80), siRNA MV (서열 번호: 81), siRNA G8 (서열 번호: 92), 또는 siRNA MF (서열 번호: 93), 또는 siRNA TV (서열 번호: 2), siRNA TU (서열 번호: 3), siRNA CL (서열 번호: 30), siRNA CM (서열 번호: 31), siRNA CR (서열 번호: 36), siRNA CV (서열 번호: 40), siRNA Y4 (서열 번호: 59), siRNA MP (서열 번호: 76), siRNA MW (서열 번호: 80), siRNA MV (서열 번호: 81), siRNA G8 (서열 번호: 92), 또는 siRNA MF (서열 번호: 93)에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO에서 선택된다. 추가적으로, 서열 번호: 116 또는 서열 번호: 115의 엑손 3의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 적어도 15% (예를 들면, 적어도 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낼 수 있다. 또 다른 구체예에서, Grik2 ASO는 siRNA TV (서열 번호: 2), siRNA TU (서열 번호: 3), siRNA CL (서열 번호: 30), siRNA CM (서열 번호: 31), siRNA CR (서열 번호: 36), siRNA CV (서열 번호: 40), siRNA Y4 (서열 번호: 59), siRNA MP (서열 번호: 76), siRNA MW (서열 번호: 80), siRNA MV (서열 번호: 81), siRNA G8 (서열 번호: 92), 또는 siRNA MF (서열 번호: 93), 또는 여기에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO에서 선택되고, 그리고 15% 초과 (예를 들면, 적어도 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다.
게다가, 개시된 ASO 작용제는 Grik2 mRNA의 엑손 4, 예컨대 예를 들면, 서열 번호: 115의 뉴클레오티드 위치 835-1016에 위치된 Grik2 mRNA의 엑손 4의 적어도 일부 또는 영역 내에 혼성화할 수 있다. Grik2 mRNA의 엑손 4의 서열은 서열 번호: 132의 핵산 서열 또는 표 4에서 도시된 바와 같은, 서열 번호: 132의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
예를 들면, 본원에서 개시된 ASO 작용제를 이용한 표적화에 대해 예기된 Grik2 표적 핵산은 서열 번호: 116의 뉴클레오티드 534-554 또는 서열 번호: 115의 뉴클레오티드 827-847, 서열 번호: 116의 뉴클레오티드 579-599 또는 서열 번호: 115의 뉴클레오티드 872-892, 서열 번호: 116의 뉴클레오티드 717-737 또는 서열 번호: 115의 뉴클레오티드 1010-1030, 서열 번호: 116의 뉴클레오티드 721-741 또는 서열 번호: 115의 뉴클레오티드 1014-1034, 서열 번호: 116의 뉴클레오티드 559-579 또는 서열 번호: 115의 뉴클레오티드 852-872, 또는 이들의 단편 또는 일부를 포함한다.
서열 번호: 116 또는 서열 번호: 115의 엑손 4의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 siRNA CV (서열 번호: 40), siRNA Y5 (서열 번호: 60), siRNA G9 (서열 번호: 68), siRNA MD (서열 번호: 70), 또는 siRNA MK (서열 번호: 86), 또는 siRNA CV (서열 번호: 40), siRNA Y5 (서열 번호: 60), siRNA G9 (서열 번호: 68), siRNA MD (서열 번호: 70), 또는 siRNA MK (서열 번호: 86)에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO에서 선택된다. 추가적으로, 서열 번호: 116 또는 서열 번호: 115의 엑손 4의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 25% 초과 (예를 들면, 적어도 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다. 더 나아가, Grik2 ASO는 siRNA CV (서열 번호: 40), siRNA Y5 (서열 번호: 60), siRNA G9 (서열 번호: 68), siRNA MD (서열 번호: 70), 또는 siRNA MK (서열 번호: 86), 또는 여기에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO에서 선택되고, 그리고 25% 초과 (예를 들면, 적어도 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다.
추가적으로, ASO 작용제는 Grik2 mRNA의 엑손 5, 예컨대 예를 들면, 서열 번호: 115의 뉴클레오티드 위치 1017-1070에 위치된 Grik2 mRNA의 엑손 5의 적어도 일부 또는 영역 내에 혼성화할 수 있다. Grik2 mRNA의 엑손 5의 서열은 서열 번호: 133의 핵산 서열 또는 표 4에서 도시된 바와 같은, 서열 번호: 133의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
예를 들면, 본원에서 개시된 ASO 작용제를 이용한 표적화에 대해 예기된 Grik2 표적 핵산은 서열 번호: 116의 뉴클레오티드 717-737 또는 서열 번호: 115의 뉴클레오티드 1010-1030, 서열 번호: 116의 뉴클레오티드 728-747 또는 서열 번호: 115의 뉴클레오티드 1021-1040, 서열 번호: 116의 뉴클레오티드 721-741 또는 서열 번호: 115의 뉴클레오티드 1014-1034, 또는 이들의 단편 또는 일부를 포함한다.
서열 번호: 116 또는 서열 번호: 115의 엑손 5의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 siRNA G9 (서열 번호: 68), siRNA ME (서열 번호: 69), 또는 siRNA MD (서열 번호: 70), 또는 siRNA G9 (서열 번호: 68), siRNA ME (서열 번호: 69), 또는 siRNA MD (서열 번호: 70)에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO에서 선택된다. 추가적으로, 서열 번호: 116 또는 서열 번호: 115의 엑손 5의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 75% 초과 (예를 들면, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 GluK2 녹다운을 나타낸다. 더 나아가, Grik2 ASO는 siRNA G9 (서열 번호: 68), siRNA ME (서열 번호: 69), 또는 siRNA MD (서열 번호: 70), 또는 여기에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO에서 선택되고, 그리고 75% 초과 (예를 들면, 적어도 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 GluK2 녹다운을 나타낸다.
ASO 작용제는 또한, Grik2 mRNA의 엑손 6, 예컨대 예를 들면, 서열 번호: 115의 뉴클레오티드 위치 1071-1244에 위치된 Grik2 mRNA의 엑손 6의 적어도 일부 또는 영역 내에 혼성화할 수 있다. Grik2 mRNA의 엑손 6의 서열은 서열 번호: 134의 핵산 서열 또는 표 4에서 도시된 바와 같은, 서열 번호: 134의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
예를 들면, 본원에서 개시된 ASO 작용제를 이용한 표적화에 대해 예기된 Grik2 표적 핵산은 서열 번호: 116의 뉴클레오티드 806-826 또는 서열 번호: 115의 뉴클레오티드 1099-1119, 서열 번호: 116의 뉴클레오티드 905-925 또는 서열 번호: 115의 뉴클레오티드 1198-1218, 서열 번호: 116의 뉴클레오티드 904-924 또는 서열 번호: 115의 뉴클레오티드 1197-1217, 서열 번호: 116의 뉴클레오티드 885-905 또는 서열 번호: 115의 뉴클레오티드 1178-1198, 서열 번호: 116의 뉴클레오티드 908-927 또는 서열 번호: 115의 뉴클레오티드 1201-1220, 서열 번호: 116의 뉴클레오티드 908-928 또는 서열 번호: 115의 뉴클레오티드 1201-1221, 서열 번호: 116의 뉴클레오티드 934-954 또는 서열 번호: 115의 뉴클레오티드 1227-1247, 서열 번호: 116의 뉴클레오티드 931-950 또는 서열 번호: 115의 뉴클레오티드 1224-1243, 서열 번호: 116의 뉴클레오티드 938-957 또는 서열 번호: 115의 뉴클레오티드 1231-1250, 또는 이들의 단편 또는 일부를 포함한다.
서열 번호: 116 또는 서열 번호: 115의 엑손 6의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 siRNA TT (서열 번호: 4), siRNA G1 (서열 번호: 5), siRNA G2 (서열 번호: 6), siRNA Y1 (서열 번호: 56), siRNA Y2 (서열 번호: 57), siRNA Y3 (서열 번호: 58), siRNA GG (서열 번호: 91), siRNA MH (서열 번호: 94), 또는 siRNA MG (서열 번호: 95), 또는 siRNA TT (서열 번호: 4), siRNA G1 (서열 번호: 5), siRNA G2 (서열 번호: 6), siRNA Y1 (서열 번호: 56), siRNA Y2 (서열 번호: 57), siRNA Y3 (서열 번호: 58), siRNA GG (서열 번호: 91), siRNA MH (서열 번호: 94), 또는 siRNA MG (서열 번호: 95)에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO에서 선택된다. 추가적으로, 서열 번호: 116 또는 서열 번호: 115의 엑손 6의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 20% 초과 (예를 들면, 적어도 20%, 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다. 또 다른 구체예에서, Grik2 ASO는 siRNA TT (서열 번호: 4), siRNA G1 (서열 번호: 5), siRNA G2 (서열 번호: 6), siRNA Y1 (서열 번호: 56), siRNA Y2 (서열 번호: 57), siRNA Y3 (서열 번호: 58), siRNA GG (서열 번호: 91), siRNA MH (서열 번호: 94), 또는 siRNA MG (서열 번호: 95), 또는 여기에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO에서 선택되고, 그리고 20% 초과 (예를 들면, 적어도 20%, 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다.
이에 더하여, ASO 작용제는 Grik2 mRNA의 엑손 7, 예컨대 예를 들면, 서열 번호: 115의 뉴클레오티드 위치 1245-1388에 위치된 Grik2 mRNA의 엑손 7의 적어도 일부 또는 영역 내에 혼성화할 수 있다. Grik2 mRNA의 엑손 7의 서열은 서열 번호: 135의 핵산 서열 또는 표 4에서 도시된 바와 같은, 서열 번호: 135의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
예를 들면, 본원에서 개시된 ASO 작용제를 이용한 표적화에 대해 예기된 Grik2 표적 핵산은 서열 번호: 116의 뉴클레오티드 1029-1049 또는 서열 번호: 115의 뉴클레오티드 1322-1342, 서열 번호: 116의 뉴클레오티드 985-1005 또는 서열 번호: 115의 뉴클레오티드 1278-1298, 서열 번호: 116의 뉴클레오티드 1057-1077 또는 서열 번호: 115의 뉴클레오티드 1350-1370, 서열 번호: 116의 뉴클레오티드 1058-1078 또는 서열 번호: 115의 뉴클레오티드 1351-1371, 서열 번호: 116의 뉴클레오티드 1043-1063 또는 서열 번호: 115의 뉴클레오티드 1336-1356, 또는 이들의 단편 또는 일부를 포함한다.
서열 번호: 116 또는 서열 번호: 115의 엑손 7의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 siRNA TL (서열 번호: 20), siRNA CS (서열 번호: 37), siRNA CT (서열 번호: 38), siRNA CZ (서열 번호: 44), 또는 siRNA D2 (서열 번호: 47), 또는 siRNA TL (서열 번호: 20), siRNA CS (서열 번호: 37), siRNA CT (서열 번호: 38), siRNA CZ (서열 번호: 44), 또는 siRNA D2 (서열 번호: 47)에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO에서 선택된다. 추가적으로, 서열 번호: 116 또는 서열 번호: 115의 엑손 7의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 45% 초과 (예를 들면, 적어도 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다. 더 나아가, Grik2 ASO는 siRNA TL (서열 번호: 20), siRNA CS (서열 번호: 37), siRNA CT (서열 번호: 38), siRNA CZ (서열 번호: 44), 또는 siRNA D2 (서열 번호: 47), 또는 여기에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO에서 선택되고, 그리고 45% 초과 (예를 들면, 적어도 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다.
ASO 작용제는 Grik2 mRNA의 엑손 8, 예컨대 예를 들면, 서열 번호: 115의 뉴클레오티드 위치 1389-1496에 위치된 Grik2 mRNA의 엑손 8의 적어도 일부 또는 영역 내에 더욱 혼성화할 수 있다. Grik2 mRNA의 엑손 8의 서열은 서열 번호: 136의 핵산 서열 또는 표 4에서 도시된 바와 같은, 서열 번호: 136의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다. 엑손 8의 일부 또는 영역을 표적으로 하는 ASO 작용제는 적어도 10% (예를 들면, 적어도 10%, 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낼 수 있다.
게다가, ASO 작용제는 Grik2 mRNA의 엑손 9, 예컨대 예를 들면, 서열 번호: 115의 뉴클레오티드 위치 1497-1610에 위치된 Grik2 mRNA의 엑손 9의 적어도 일부 또는 영역 내에 혼성화할 수 있다. Grik2 mRNA의 엑손 9의 서열은 서열 번호: 137의 핵산 서열 또는 표 4에서 도시된 바와 같은, 서열 번호: 137의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
예를 들면, 본원에서 개시된 ASO 작용제를 이용한 표적화에 대해 예기된 Grik2 표적 핵산은 서열 번호: 116의 뉴클레오티드 1252-1272 또는 서열 번호: 115의 뉴클레오티드 1545-1565, 또는 이들의 단편 또는 일부를 포함한다.
서열 번호: 116 또는 서열 번호: 115의 엑손 9의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 siRNA TQ (서열 번호: 12), 또는 siRNA TQ (서열 번호: 12)에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO이다. 추가적으로, 서열 번호: 116 또는 서열 번호: 115의 엑손 9의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 50% 초과 (예를 들면, 적어도 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다. 더 나아가, Grik2 ASO는 siRNA TQ (서열 번호: 12), 또는 여기에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO이고, 그리고 50% 초과 (예를 들면, 적어도 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다.
ASO 작용제는 Grik2 mRNA의 엑손 10, 예컨대 예를 들면, 서열 번호: 115의 뉴클레오티드 위치 1611-1817에 위치된 Grik2 mRNA의 엑손 10에 추가적으로 혼성화할 수 있다. Grik2 mRNA의 엑손 10의 서열은 서열 번호: 138의 핵산 서열 또는 표 4에서 도시된 바와 같은, 서열 번호: 138의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
예를 들면, 본원에서 개시된 ASO 작용제를 이용한 표적화에 대해 예기된 Grik2 표적 핵산은 서열 번호: 116의 뉴클레오티드 1396-1416 또는 서열 번호: 115의 뉴클레오티드 1689-1709, 서열 번호: 116의 뉴클레오티드 1496-1516 또는 서열 번호: 115의 뉴클레오티드 1789-1809, 서열 번호: 116의 뉴클레오티드 1417-1437 또는 서열 번호: 115의 뉴클레오티드 1710-1730, 서열 번호: 116의 뉴클레오티드 1483-1503 또는 서열 번호: 115의 뉴클레오티드 1776-1796, 서열 번호: 116의 뉴클레오티드 1491-1511 또는 서열 번호: 115의 뉴클레오티드 1784-1804, 또는 이들의 단편 또는 일부를 포함한다.
서열 번호: 116 또는 서열 번호: 115의 엑손 10의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 siRNA GD (서열 번호: 7), G3 (서열 번호: 8), siRNA MU (서열 번호: 96), siRNA MT (서열 번호: 98), 또는 siRNA MS (서열 번호: 99), 또는 siRNA GD (서열 번호: 7), G3 (서열 번호: 8), siRNA MU (서열 번호: 96), siRNA MT (서열 번호: 98), 또는 siRNA MS (서열 번호: 99)에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO에서 선택된다. 추가적으로, 서열 번호: 116 또는 서열 번호: 115의 엑손 10의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 50% 초과 (예를 들면, 적어도 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다. 더 나아가, Grik2 ASO는 GD (서열 번호: 7), G3 (서열 번호: 8), siRNA MU (서열 번호: 96), siRNA MT (서열 번호: 98), 또는 siRNA MS (서열 번호: 99), 또는 여기에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO에서 선택되고, 그리고 50% 초과 (예를 들면, 적어도 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다.
게다가, ASO 작용제는 Grik2 mRNA의 엑손 11, 예컨대 예를 들면, 서열 번호: 115의 뉴클레오티드 위치 1818-2041에 위치된 Grik2 mRNA의 엑손 11의 적어도 일부 또는 영역 내에 혼성화할 수 있다. Grik2 mRNA의 엑손 11의 서열은 서열 번호: 139의 핵산 서열 또는 표 4에서 도시된 바와 같은, 서열 번호: 139의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
예를 들면, 본원에서 개시된 ASO 작용제를 이용한 표적화에 대해 예기된 Grik2 표적 핵산은 서열 번호: 116의 뉴클레오티드 1550-1570 또는 서열 번호: 115의 뉴클레오티드 1843-1863, 서열 번호: 116의 뉴클레오티드 1637-1657 또는 서열 번호: 115의 뉴클레오티드 1930-1950, 서열 번호: 116의 뉴클레오티드 1670-1690 또는 서열 번호: 115의 뉴클레오티드 1963-1983, 서열 번호: 116의 뉴클레오티드 1565-1585 또는 서열 번호: 115의 뉴클레오티드 1858-1878, 서열 번호: 116의 뉴클레오티드 1550-1569 또는 서열 번호: 115의 뉴클레오티드 1843-1862, 서열 번호: 116의 뉴클레오티드 1544-1563 또는 서열 번호: 115의 뉴클레오티드 1837-1856, 서열 번호: 116의 뉴클레오티드 1544-1564 또는 서열 번호: 115의 뉴클레오티드 1837-1857, 서열 번호: 116의 뉴클레오티드 1526-1546, 서열 번호: 116의 뉴클레오티드 1541-1561 또는 서열 번호: 115의 뉴클레오티드 1834-1854, 또는 이들의 단편 또는 일부를 포함한다.
서열 번호: 116 또는 서열 번호: 115의 엑손 11의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 siRNA TH (서열 번호: 22), siRNA CU (서열 번호: 39), siRNA Y7 (서열 번호: 62), siRNA TK (서열 번호: 74), siRNA TI (서열 번호: 75), siRNA Y8 (서열 번호: 87), siRNA Y9 (서열 번호: 88), siRNA MJ (서열 번호: 89), 또는 siRNA MI (서열 번호: 90), 또는 siRNA TH (서열 번호: 22), siRNA CU (서열 번호: 39), siRNA Y7 (서열 번호: 62), siRNA TK (서열 번호: 74), siRNA TI (서열 번호: 75), siRNA Y8 (서열 번호: 87), siRNA Y9 (서열 번호: 88), siRNA MJ (서열 번호: 89), 또는 siRNA MI (서열 번호: 90)에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO에서 선택된다. 추가적으로, 서열 번호: 116 또는 서열 번호: 115의 엑손 11의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 25% 초과 (예를 들면, 적어도 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다. 더 나아가, Grik2 ASO는 siRNA TH (서열 번호: 22), siRNA CU (서열 번호: 39), siRNA Y7 (서열 번호: 62), siRNA TK (서열 번호: 74), siRNA TI (서열 번호: 75), siRNA Y8 (서열 번호: 87), siRNA Y9 (서열 번호: 88), siRNA MJ (서열 번호: 89), 또는 siRNA MI (서열 번호: 90), 또는 여기에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO에서 선택되고, 그리고 25% 초과 (예를 들면, 적어도 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다.
추가 실례로서, ASO 작용제는 Grik2 mRNA의 엑손 12, 예컨대 예를 들면, 서열 번호: 115의 뉴클레오티드 위치 2042-2160에 위치된 Grik2 mRNA의 엑손 12의 적어도 일부 또는 영역 내에 혼성화할 수 있다. Grik2 mRNA의 엑손 12의 서열은 서열 번호: 140의 핵산 서열 또는 표 4에서 도시된 바와 같은, 서열 번호: 140의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
예를 들면, 본원에서 개시된 ASO 작용제를 이용한 표적화에 대해 예기된 Grik2 표적 핵산은 서열 번호: 116의 뉴클레오티드 1786-1805 또는 서열 번호: 115의 뉴클레오티드 2079-2098, 서열 번호: 116의 뉴클레오티드 1786-1806 또는 서열 번호: 115의 뉴클레오티드 2079-2099, 서열 번호: 116의 뉴클레오티드 1778-1797 또는 서열 번호: 115의 뉴클레오티드 2071-2090, 서열 번호: 116의 뉴클레오티드 1836-1856 또는 서열 번호: 115의 뉴클레오티드 2129-2149, 또는 이들의 단편 또는 일부를 포함한다.
서열 번호: 116 또는 서열 번호: 115의 엑손 12의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 siRNA XX (서열 번호: 82), siRNA XY (서열 번호: 83), siRNA MM (서열 번호: 84), 또는 siRNA ML (서열 번호: 85), 또는 siRNA XX (서열 번호: 82), siRNA XY (서열 번호: 83), siRNA MM (서열 번호: 84), 또는 siRNA ML (서열 번호: 85)에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO에서 선택된다. 추가적으로, 서열 번호: 116 또는 서열 번호: 115의 엑손 12의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 50% 초과 (예를 들면, 적어도 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다. 더 나아가, Grik2 ASO는 siRNA XX (서열 번호: 82), siRNA XY (서열 번호: 83), siRNA MM (서열 번호: 84), 또는 siRNA ML (서열 번호: 85), 또는 여기에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO에서 선택되고, 그리고 50% 초과 (예를 들면, 적어도 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다.
ASO 작용제는 또한, Grik2 mRNA의 엑손 13, 예컨대 예를 들면, 서열 번호: 115의 뉴클레오티드 위치 2161-2378에 위치된 Grik2 mRNA의 엑손 13의 적어도 일부 또는 영역 내에 혼성화할 수 있다. Grik2 mRNA의 엑손 13의 서열은 서열 번호: 141의 핵산 서열 또는 표 4에서 도시된 바와 같은, 서열 번호: 141의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
예를 들면, 본원에서 개시된 ASO 작용제를 이용한 표적화에 대해 예기된 Grik2 표적 핵산은 서열 번호: 116의 뉴클레오티드 1968-1987 또는 서열 번호: 115의 뉴클레오티드 2213-2233, 서열 번호: 116의 뉴클레오티드 1968-1988 또는 서열 번호: 115의 뉴클레오티드 2213-2233, 서열 번호: 116의 뉴클레오티드 1906-1926 또는 서열 번호: 115의 뉴클레오티드 2199-2219, 서열 번호: 116의 뉴클레오티드 1920-1940 또는 서열 번호: 115의 뉴클레오티드 2213-2233, 또는 이들의 단편 또는 일부를 포함한다.
서열 번호: 116 또는 서열 번호: 115의 엑손 13의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 siRNA TP (서열 번호: 13), siRNA TO (서열 번호: 14), siRNA MR (서열 번호: 72), 또는 siRNA MQ (서열 번호: 73), 또는 siRNA TP (서열 번호: 13), siRNA TO (서열 번호: 14), siRNA MR (서열 번호: 72), 또는 siRNA MQ (서열 번호: 73)에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO에서 선택된다. 추가적으로, 서열 번호: 116 또는 서열 번호: 115의 엑손 13의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 35% 초과 (예를 들면, 적어도 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다. 더 나아가, Grik2 ASO는 siRNA TP (서열 번호: 13), siRNA TO (서열 번호: 14), siRNA MR (서열 번호: 72), 또는 siRNA MQ (서열 번호: 73), 또는 여기에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO에서 선택되고, 그리고 35% 초과 (예를 들면, 적어도 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다.
추가적으로, ASO 작용제는 Grik2 mRNA의 엑손 14, 예컨대 예를 들면, 서열 번호: 115의 뉴클레오티드 위치 2379-2604에 위치된 Grik2 mRNA의 엑손 14의 적어도 일부 또는 영역 내에 혼성화할 수 있다. Grik2 mRNA의 엑손 14의 서열은 서열 번호: 142의 핵산 서열 또는 표 4에서 도시된 바와 같은, 서열 번호: 142의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
예를 들면, 본원에서 개시된 ASO 작용제를 이용한 표적화에 대해 예기된 Grik2 표적 핵산은 서열 번호: 116의 뉴클레오티드 2209-2228 또는 서열 번호: 115의 뉴클레오티드 2502-2521, 서열 번호: 116의 뉴클레오티드 2209-2229 또는 서열 번호: 115의 뉴클레오티드 2502-2522, 서열 번호: 116의 뉴클레오티드 2308-2328 또는 서열 번호: 115의 뉴클레오티드 2601-2621, 서열 번호: 116의 뉴클레오티드 2304-2323 또는 서열 번호: 115의 뉴클레오티드 2597-2616, 서열 번호: 116의 뉴클레오티드 2303-2323 또는 서열 번호: 115의 뉴클레오티드 2596-2616, 또는 이들의 단편 또는 일부를 포함한다.
서열 번호: 116 또는 서열 번호: 115의 엑손 14의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 siRNA CP (서열 번호: 34), siRNA CQ (서열 번호: 35), siRNA GI (서열 번호: 77), siRNA MO (서열 번호: 78), 또는 siRNA MN (서열 번호: 79), 또는 siRNA CP (서열 번호: 34), siRNA CQ (서열 번호: 35), siRNA GI (서열 번호: 77), siRNA MO (서열 번호: 78), 또는 siRNA MN (서열 번호: 79)에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO에서 선택된다. 추가적으로, 서열 번호: 116 또는 서열 번호: 115의 엑손 14의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 35% 초과 (예를 들면, 적어도 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다. 더 나아가, Grik2 ASO는 siRNA CP (서열 번호: 34), siRNA CQ (서열 번호: 35), siRNA GI (서열 번호: 77), siRNA MO (서열 번호: 78), 또는 siRNA MN (서열 번호: 79), 또는 여기에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO에서 선택되고, 그리고 35% 초과 (예를 들면, 적어도 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다.
ASO 작용제는 또한, Grik2 mRNA의 엑손 15, 예컨대 예를 들면, 서열 번호: 115의 뉴클레오티드 위치 2605-2855에 위치된 Grik2 mRNA의 엑손 15의 적어도 일부 또는 영역 내에 혼성화할 수 있다. Grik2 mRNA의 엑손 15의 뉴클레오티드 서열은 서열 번호: 143의 핵산 서열 또는 표 4에서 도시된 바와 같은, 서열 번호: 143의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
예를 들면, 본원에서 개시된 ASO 작용제를 이용한 표적화에 대해 예기된 Grik2 표적 핵산은 서열 번호: 116의 뉴클레오티드 2309-2329 또는 서열 번호: 115의 뉴클레오티드 2602-2622, 또는 이들의 단편 또는 일부를 포함한다.
서열 번호: 116 또는 서열 번호: 115의 엑손 15의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 siRNA XU (서열 번호: 51), 또는 siRNA XU (서열 번호: X)에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO이다. 추가적으로, 서열 번호: 116 또는 서열 번호: 115의 엑손 15의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 50% 초과 (예를 들면, 적어도 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다. 더 나아가, Grik2 ASO는 siRNA XU (서열 번호: 51), 또는 여기에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO이고, 그리고 50% 초과 (예를 들면, 적어도 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다.
게다가, ASO 작용제는 Grik2 mRNA의 엑손 16, 예컨대 예를 들면, 서열 번호: 115의 뉴클레오티드 위치 2856-4592에 위치된 Grik2 mRNA의 엑손 16의 적어도 일부 또는 영역 내에 혼성화할 수 있다. Grik2 mRNA의 엑손 16의 서열은 서열 번호: 144의 핵산 서열 또는 표 4에서 도시된 바와 같은, 서열 번호: 144의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
예를 들면, 본원에서 개시된 ASO 작용제를 이용한 표적화에 대해 예기된 Grik2 표적 핵산은 서열 번호: 116의 뉴클레오티드 2632-2652 또는 서열 번호: 115의 뉴클레오티드 2925-2945, 서열 번호: 115의 뉴클레오티드 3382-3402, 서열 번호: 115의 뉴클레오티드 3792-3812, 서열 번호: 115의 뉴클레오티드 3347-3367, 서열 번호: 115의 뉴클레오티드 3605-3625, 서열 번호: 116의 뉴클레오티드 2581-2601 또는 서열 번호: 115의 뉴클레오티드 2874-2893, 서열 번호: 116의 뉴클레오티드 2581-2601 또는 서열 번호: 115의 뉴클레오티드 2874-2893, 서열 번호: 115의 뉴클레오티드 4289-4309, 서열 번호: 115의 뉴클레오티드 4274-4293, 서열 번호: 115의 뉴클레오티드 4274-4294, 서열 번호: 115의 뉴클레오티드 4078-4098, 서열 번호: 115의 뉴클레오티드 3037-3057, 서열 번호: 115의 뉴클레오티드 4417-4437, 서열 번호: 116의 뉴클레오티드 2601-2620 또는 서열 번호: 115의 뉴클레오티드 2894-2913, 서열 번호: 116의 뉴클레오티드 2601-2621 또는 서열 번호: 115의 뉴클레오티드 2894-2914, 서열 번호: 115의 뉴클레오티드 3479-3499, 서열 번호: 115의 뉴클레오티드 3085-3105, 또는 이들의 단편 또는 일부를 포함한다.
서열 번호: 116 또는 서열 번호: 115의 엑손 16의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 siRNA G4 (서열 번호: 9), siRNA TS (서열 번호: 10), siRNA TR (서열 번호: 11), siRNA G5 (서열 번호: 15), siRNA TN (서열 번호: 16), siRNA G6 (서열 번호: 18), siRNA G7 (서열 번호: 19), siRNA GJ (서열 번호: 27), siRNA CN (서열 번호: 32), siRNA CO (서열 번호: 33), siRNA CW (서열 번호: 41), siRNA XS (서열 번호: 49), siRNA XT (서열 번호: 50), siRNA XV (서열 번호: 52), siRNA XW (서열 번호: 53), siRNA Y6 (서열 번호: 61), 또는 siRNA YA (서열 번호: 63), 또는 siRNA G4 (서열 번호: 9), siRNA TS (서열 번호: 10), siRNA TR (서열 번호: 11), siRNA G5 (서열 번호: 15), siRNA TN (서열 번호: 16), siRNA G6 (서열 번호: 18), siRNA G7 (서열 번호: 19), siRNA GJ (서열 번호: 27), siRNA CN (서열 번호: 32), siRNA CO (서열 번호: 33), siRNA CW (서열 번호: 41), siRNA XS (서열 번호: 49), siRNA XT (서열 번호: 50), siRNA XV (서열 번호: 52), siRNA XW (서열 번호: 53), siRNA Y6 (서열 번호: 61), 또는 siRNA YA (서열 번호: 63)에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO에서 선택된다. 추가적으로, 서열 번호: 116 또는 서열 번호: 115의 엑손 16의 일부 또는 영역 내에 핵산을 표적으로 하는 Grik2 ASO는 5% 초과 (예를 들면, 적어도 5%, 10%, 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다. 더 나아가, Grik2 ASO는 siRNA G4 (서열 번호: 9), siRNA TS (서열 번호: 10), siRNA TR (서열 번호: 11), siRNA G5 (서열 번호: 15), siRNA TN (서열 번호: 16), siRNA G6 (서열 번호: 18), siRNA G7 (서열 번호: 19), siRNA GJ (서열 번호: 27), siRNA CN (서열 번호: 32), siRNA CO (서열 번호: 33), siRNA CW (서열 번호: 41), siRNA XS (서열 번호: 49), siRNA XT (서열 번호: 50), siRNA XV (서열 번호: 52), siRNA XW (서열 번호: 53), siRNA Y6 (서열 번호: 61), 또는 siRNA YA (서열 번호: 63), 또는 여기에 85% 초과 (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 ASO에서 선택되고, 그리고 5% 초과 (예를 들면, 적어도 5%, 10%, 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 GluK2 녹다운을 나타낸다.
안티센스 올리고뉴클레오티드 및 Grik2 표적 영역의 열역학적 특성
RNA 이차 구조, 예컨대 예를 들면, 본원 발명의 안티센스 작용제 또는 이들이 혼성화하는 표적 서열 (예를 들면, Grik2 표적 서열)의 상응하는 영역에 의해 형성된 것들은 열역학으로부터 차용된 개념, 예컨대 엔트로피 및 열역학적 자유 에너지를 이용하여 설명될 수 있다. 열역학적 자유 에너지는 일반적으로, 시스템이 일정한 온도에서 과정 동안 수행할 수 있는 최대량의 작업으로서 설명되고, 그리고 상기 과정이 열역학적으로 호의적인지 또는 금지적인지를 나타낸다. 간단히 말하면, 열역학적 자유 에너지는 물리적 상태에서 변화를 겪는 시스템의 능력을 지칭한다. 폴리뉴클레오티드 (예를 들면, 본원 발명의 ASO 작용제 또는 이의 실제적으로 상보성 서열)의 맥락에서, 열역학적 자유 에너지에 기초된 측정항목은 특정 이차 구조가 분해될 수 있는 용이함 (다시 말하면, 안티센스 올리고뉴클레오티드 또는 이의 부분적인 또는 완전한 보체 중 어느 한 가지의 이차 RNA 구조를 개방하는 데 필요한 에너지), RNA 분자 사이에 또는 내에 이중나선 형성으로부터 생성된 에너지, 그리고 각 RNA를 분해하는 데 필요한 총 에너지 및 그 자체로 혼성화의 에너지를 고려하는, 그 자체 또는 다른 RNA 분자에 대한 RNA 분자의 총 결합 에너지를 설명할 수 있다.
본원 발명은 RNA 분자 (예를 들면, 본원 발명의 ASO 작제물 또는 이들의 실제적으로 상보성 서열, 예컨대 예를 들면, Grik2 표적 영역)의 열역학적 특징이, 안티센스 분자가 표적 mRNA의 발현을 녹다운시킬 수 있는 효능을 예측하는 데 이용될 수 있다는, 본 발명자들에 의해 이루어진 발견에 부분적으로 근거된다. 따라서, 본원에서 개시된 조성물과 방법은 mRNA 발현의 녹다운의 가능성을 예측하기 위해 열역학적 파라미터를 이용하여 ASO 서열 또는 이의 표적 mRNA 서열을 특징화할 수 있다.
특히, 본원 발명은 표적 mRNA 영역에 대하여 특정 ASO 서열의 녹다운 효능을 예측하는 데 유용한 3가지 구별되는 열역학적 파라미터, 다시 말하면 결합의 총 자유 에너지, 이중나선 형성으로부터 에너지, 그리고 표적 개방 에너지 (또는 개방 에너지)를 제공한다. RNA 분자의 열역학적 안정성을 특징화하고 특정 ASO 작용제의 녹다운 효능을 예측하는 데 이용될 수 있는 추가의 개념은 RNA 분자의 GC (구아닌-시토신; %) 함량이다. 본원 발명의 맥락에서, ASO의 결합의 총 자유 에너지 (kcal/mol)는 상응하는 표적 mRNA 서열에 혼성화하는 ASO의 과정의 자유 에너지를 지칭한다. 여기에는 mRNA (예를 들면, Grik2 mRNA)의 표적 영역을 개방하는 데 필요한 에너지, 단일 가닥 안티센스 가이드 서열을 생성하는 데 필요한 에너지, 그리고 폴리뉴클레오티드 및 이의 보체 사이에 혼성화 (완전 또는 실제적)의 에너지가 포함된다. 이와 관련하여, 이중나선 형성으로부터 에너지는 2개의 RNA 분자 사이에 이중나선 구조의 형성의 호감도 및 결과로서, RNA 이중나선의 안정성을 표시하는 열역학적 특성을 지칭한다. 총 개방 에너지는 인근 이차 구조의 분해 또는 표적 서열과 이차 구조를 형성하는 원위 서열의 수반을 비롯하여, 표적 위치에서 RNA 이차 구조를 분해하는 (다시 말하면, 개방하는/접근 가능하게 만드는) 데 필요한 에너지를 반영하는 열역학적 측정항목이다.
따라서, 본원 발명은 10 kcal/mol보다 작은 (예를 들면, 10 kcal/mol, 9 kcal/mol, 8 kcal/mol, 7 kcal/mol, 6 kcal/mol, 5 kcal/mol, 4 kcal/mol, 3 kcal/mol, 2 kcal/mol, 또는 1 kcal/mol보다 작은) 총 개방 에너지를 갖는 ASO 서열 (예컨대 예를 들면, 본원에서 개시된 ASO 서열)을 예기한다. 특정 실례에서, 본원 발명의 ASO 서열은 9 kcal/mol보다 작은 (예를 들면, 8 kcal/mol, 7 kcal/mol, 6 kcal/mol, 5 kcal/mol, 4 kcal/mol, 3 kcal/mol, 2 kcal/mol, 또는 1 kcal/mol, 또는 그 미만보다 작은) 총 개방 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 8 kcal/mol 보다 작은 (예를 들면, 7 kcal/mol, 6 kcal/mol, 5 kcal/mol, 4 kcal/mol, 3 kcal/mol, 2 kcal/mol, 또는 1 kcal/mol, 또는 그 미만보다 작은) 총 개방 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 7 kcal/mol보다 작은 (예를 들면, 6 kcal/mol, 5 kcal/mol, 4 kcal/mol, 3 kcal/mol, 2 kcal/mol, 또는 1 kcal/mol, 또는 그 미만보다 작은) 총 개방 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 6 kcal/mol보다 작은 (예를 들면, 5 kcal/mol, 4 kcal/mol, 3 kcal/mol, 2 kcal/mol, 또는 1 kcal/mol, 또는 그 미만보다 작은) 총 개방 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 5 kcal/mol보다 작은 (예를 들면, 4 kcal/mol, 3 kcal/mol, 2 kcal/mol, 또는 1 kcal/mol, 또는 그 미만보다 작은) 총 개방 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 4 kcal/mol보다 작은 (예를 들면, 3 kcal/mol, 2 kcal/mol, 또는 1 kcal/mol, 또는 그 미만보다 작은) 총 개방 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 3 kcal/mol보다 작은 (예를 들면, 2 kcal/mol, 또는 1 kcal/mol, 또는 그 미만보다 작은) 총 개방 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 2 kcal/mol보다 작은 (예를 들면, 1 kcal/mol, 또는 그 미만) 총 개방 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 1 kcal/mol보다 작은 총 개방 에너지를 갖는다.
게다가, -41 kcal/mol보다 큰 (예를 들면, -40 kcal/mol, -38 kcal/mol, -35 kcal/mol, -30 kcal/mol, -25 kcal/mol, -20 kcal/mol, -15 kcal/mol, -10 kcal/mol, -5 kcal/mol, -4 kcal/mol, -3 kcal/mol, -2 kcal/mol, -1 kcal/mol 또는 그 초과보다 큰) 이중나선 형성의/으로부터 에너지를 갖는 ASO 서열이 본원에서 개시된다. 다른 실례에서, 본원 발명의 ASO 서열은 -38 kcal/mol보다 큰 (예를 들면, -35 kcal/mol, -30 kcal/mol, -25 kcal/mol, -20 kcal/mol, -15 kcal/mol, -10 kcal/mol, -5 kcal/mol, -4 kcal/mol, -3 kcal/mol, -2 kcal/mol, -1 kcal/mol 또는 그 초과) 이중나선 형성으로부터 에너지를 갖는다. 일부 실례에서, 본원 발명의 ASO 서열은 -35 kcal/mol보다 큰 (예를 들면, -30 kcal/mol, -25 kcal/mol, -20 kcal/mol, -15 kcal/mol, -10 kcal/mol, -5 kcal/mol, -4 kcal/mol, -3 kcal/mol, -2 kcal/mol, -1 kcal/mol 또는 그 초과보다 큰) 이중나선 형성으로부터 에너지를 갖는다. 특정 실례에서, 본원 발명의 ASO 서열은 -30 kcal/mol보다 큰 (예를 들면, -25 kcal/mol, -20 kcal/mol, -15 kcal/mol, -10 kcal/mol, -5 kcal/mol, -4 kcal/mol, -3 kcal/mol, -2 kcal/mol, -1 kcal/mol 또는 그 초과보다 큰) 이중나선 형성으로부터 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 -25 kcal/mol보다 큰 (예를 들면, -20 kcal/mol, -15 kcal/mol, -10 kcal/mol, -5 kcal/mol, -4 kcal/mol, -3 kcal/mol, -2 kcal/mol, -1 kcal/mol 또는 그 초과보다 큰) 이중나선 형성으로부터 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 -20 kcal/mol보다 큰 (예를 들면, -15 kcal/mol, -10 kcal/mol, -5 kcal/mol, -4 kcal/mol, -3 kcal/mol, -2 kcal/mol, -1 kcal/mol 또는 그 초과보다 큰) 이중나선 형성으로부터 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 -15 kcal/mol보다 큰 (예를 들면, -10 kcal/mol, -5 kcal/mol, -4 kcal/mol, -3 kcal/mol, -2 kcal/mol, -1 kcal/mol 또는 그 초과보다 큰) 이중나선 형성으로부터 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 -10 kcal/mol보다 큰 (예를 들면, -5 kcal/mol, -4 kcal/mol, -3 kcal/mol, -2 kcal/mol, -1 kcal/mol 또는 그 초과보다 큰) 이중나선 형성으로부터 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 -5 kcal/mol보다 큰 (예를 들면, -4 kcal/mol, -3 kcal/mol, -2 kcal/mol, -1 kcal/mol 또는 그 초과보다 큰) 이중나선 형성으로부터 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 -4 kcal/mol보다 큰 (예를 들면, -3 kcal/mol, -2 kcal/mol, -1 kcal/mol 또는 그 초과보다 큰) 이중나선 형성으로부터 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 -3 kcal/mol보다 큰 (예를 들면, -3 kcal/mol, -2 kcal/mol, -1 kcal/mol 또는 그 초과보다 큰) 이중나선 형성으로부터 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 -2 kcal/mol보다 큰 (예를 들면, -2 kcal/mol, -1 kcal/mol 또는 그 초과보다 큰) 이중나선 형성으로부터 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 -1 kcal/mol보다 큰 이중나선 형성으로부터 에너지를 갖는다.
추가적으로, 본원 발명은 -30.5 kcal/mol보다 큰 (예를 들면, -27 kcal/mol, -24 kcal/mol, -20 kcal/mol, -15 kcal/mol, -10 kcal/mol, -5 kcal/mol, -4 kcal/mol, -3 kcal/mol, -2 kcal/mol, -1 kcal/mol, 또는 그 초과보다 큰) 결합의 총 자유 에너지를 갖는 ASO 서열에 더욱 관계한다. 일부 실례에서, 본원 발명의 ASO 서열은 -27 kcal/mol보다 큰 (예를 들면, -24 kcal/mol, -20 kcal/mol, -15 kcal/mol, -10 kcal/mol, -5 kcal/mol, -4 kcal/mol, -3 kcal/mol, -2 kcal/mol, -1 kcal/mol 또는 그 초과보다 큰) 결합의 총 자유 에너지를 갖는다. 또 다른 실례에서, 본원 발명의 ASO 서열은 -24 kcal/mol보다 큰 (예를 들면, -20 kcal/mol, -15 kcal/mol, -10 kcal/mol, -5 kcal/mol, -4 kcal/mol, -3 kcal/mol, -2 kcal/mol, -1 kcal/mol 또는 그 초과보다 큰) 결합의 총 자유 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 -20 kcal/mol보다 큰 (예를 들면, -15 kcal/mol, -10 kcal/mol, -5 kcal/mol, -4 kcal/mol, -3 kcal/mol, -2 kcal/mol, -1 kcal/mol 또는 그 초과보다 큰) 결합의 총 자유 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 -15 kcal/mol보다 큰 (예를 들면, -10 kcal/mol, -5 kcal/mol, -4 kcal/mol, -3 kcal/mol, -2 kcal/mol, -1 kcal/mol 또는 그 초과보다 큰) 결합의 총 자유 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 -10 kcal/mol보다 큰 (예를 들면, -5 kcal/mol, -4 kcal/mol, -3 kcal/mol, -2 kcal/mol, -1 kcal/mol 또는 그 초과보다 큰) 결합의 총 자유 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 -5 kcal/mol보다 큰 (예를 들면, -4 kcal/mol, -3 kcal/mol, -2 kcal/mol, -1 kcal/mol 또는 그 초과보다 큰) 결합의 총 자유 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 -4 kcal/mol보다 큰 (예를 들면, -3 kcal/mol, -2 kcal/mol, -1 kcal/mol 또는 그 초과보다 큰) 결합의 총 자유 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 -3 kcal/mol보다 큰 (예를 들면, -3 kcal/mol, -2 kcal/mol, -1 kcal/mol 또는 그 초과보다 큰) 결합의 총 자유 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 -2 kcal/mol보다 큰 (예를 들면, -2 kcal/mol, -1 kcal/mol 또는 그 초과보다 큰) 결합의 총 자유 에너지를 갖는다. 다른 실례에서, 본원 발명의 ASO 서열은 -1 kcal/mol보다 큰 결합의 총 자유 에너지를 갖는다.
게다가, 본원 발명은 또한, 60%보다 적은 (예를 들면, 55%, 50%, 45%, 40%, 35%, 30%, 25%, 20%, 15%, 10%, 5%, 4%, 3%, 2%, 1%, 또는 그 미만보다 적은) GC 함량을 갖는 ASO 서열을 예기한다. 다른 실례에서, ASO 서열은 55%보다 적은 (예를 들면, 50%, 45%, 40%, 35%, 30%, 25%, 20%, 15%, 10%, 5%, 4%, 3%, 2%, 1%, 또는 그 미만보다 적은) GC 함량을 갖는다. 또 다른 실례에서, ASO 서열은 50%보다 적은 (예를 들면, 45%, 40%, 35%, 30%, 25%, 20%, 15%, 10%, 5%, 4%, 3%, 2%, 1%, 또는 그 미만보다 적은) GC 함량을 갖는다. 특정 실례에서, ASO 서열은 45%보다 적은 (예를 들면, 40%, 35%, 30%, 25%, 20%, 15%, 10%, 5%, 4%, 3%, 2%, 1%, 또는 그 미만보다 적은) GC 함량을 갖는다. 다른 실례에서, ASO 서열은 40%보다 적은 (예를 들면, 35%, 30%, 25%, 20%, 15%, 10%, 5%, 4%, 3%, 2%, 1%, 또는 그 미만보다 적은) GC 함량을 갖는다. 다른 실례에서, ASO 서열은 35%보다 적은 (예를 들면, 30%, 25%, 20%, 15%, 10%, 5%, 4%, 3%, 2%, 1%, 또는 그 미만보다 적은) GC 함량을 갖는다. 다른 실례에서, ASO 서열은 30%보다 적은 (예를 들면, 25%, 20%, 15%, 10%, 5%, 4%, 3%, 2%, 1%, 또는 그 미만보다 적은) GC 함량을 갖는다. 다른 실례에서, ASO 서열은 35%보다 적은 (예를 들면, 30%, 25%, 20%, 15%, 10%, 5%, 4%, 3%, 2%, 1%, 또는 그 미만보다 적은) GC 함량을 갖는다. 다른 실례에서, ASO 서열은 25%보다 적은 (예를 들면, 20%, 15%, 10%, 5%, 4%, 3%, 2%, 1%, 또는 그 미만보다 적은) GC 함량을 갖는다. 다른 실례에서, ASO 서열은 20%보다 적은 (예를 들면, 15%, 10%, 5%, 4%, 3%, 2%, 1%, 또는 그 미만보다 적은) GC 함량을 갖는다. 다른 실례에서, ASO 서열은 15%보다 적은 (예를 들면, 10%, 5%, 4%, 3%, 2%, 1%, 또는 그 미만보다 적은) GC 함량을 갖는다. 다른 실례에서, ASO 서열은 10%보다 적은 (예를 들면, 5%, 4%, 3%, 2%, 1%, 또는 그 미만보다 적은) GC 함량을 갖는다. 다른 실례에서, ASO 서열은 5%보다 적은 (예를 들면, 4%, 3%, 2%, 1%, 또는 그 미만보다 적은) GC 함량을 갖는다. 다른 실례에서, ASO 서열은 4%보다 적은 (예를 들면, 3%, 2%, 1%, 또는 그 미만보다 적은) GC 함량을 갖는다. 다른 실례에서, ASO 서열은 3%보다 적은 (예를 들면, 2%, 1%, 또는 그 미만보다 적은) GC 함량을 갖는다. 다른 실례에서, ASO 서열은 2%보다 적은 (예를 들면, 1%, 또는 그 미만보다 적은) GC 함량을 갖는다. 다른 실례에서, ASO 서열은 1%보다 적은 GC 함량을 갖는다.
생체분자, 예컨대 RNA 분자 (예를 들면, 본원 발명의 ASO RNA 분자 또는 이의 실제적으로 상보성 서열)의 열역학적 특징을 결정하는 방법은 당해 분야에서 널리 알려져 있다. 예를 들면, Gruber et al. (Nucleic Acids 36:W70-4, 2008)은 RNA 서열의 설계 및 RNA 분자의 접힘과 열역학적 특징의 분석에 이용될 수 있는 도구의 수집물을 요약한다. Gruber et al.의 개시는 이것이 RNA 분자의 열역학적 특성을 결정하는 방법에 관계하는 한에 있어서, 본원에서 참조로서 편입된다.
Grik2 mRNA 이차 구조
RNA 유발 침묵 복합체 (RISC)는 짧은 간섭 RNA (siRNA)를 비롯한 단일 가닥 작은 RNA (smRNA) 및 엔도뉴클레아제로 활성 아르고노트 단백질로 구성되고, 이러한 smRNA (예를 들면, ASO, 예컨대 예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA, 또는 shmiRNA)에 상보적인 mRNA를 개열할 수 있는 리보핵산단백질 입자이다 (Pratt AJ, MacRae IJ. The RNA-induced silencing complex: a versatile gene-silencing machine. J Biol Chem.; 284(27):17897-17901, 2009; 이것은 본원에 온전히 편입된다). RISC 부하는 mRNA 녹다운의 정도를 지배하는 다양한 인자에 의해 영향을 받는 것으로 밝혀졌다. 표적 mRNA 및 안티센스 서열의 뉴클레오티드 서열은 불량한 RISC 부하, 이중나선 풀림, 그리고 감소된 특이성에 기여할 수 있다. 표적 부위 이차 구조는 smRNA-상보성과 무관하게 RISC-표적 어닐링에 영향을 줄 수 있다. 이론에 한정됨 없이, 낮은 염기 대합 확률 및/또는 높은 위치 엔트로피를 갖는 것으로 결정된 Grik2 전사체의 일정한 표적 부위 이차 구조 (도 1a와 1b의 척도에서 증가하는 강도로 음영됨; 실시예 1을 또한 참조한다)가 확인되었고 가이드 (예를 들면, 안티센스 서열) 설계를 위해 우선적으로 처리되었다. 이들 영역은 분명하게 묘사된 루프 영역 ("중심 루프," 또는 단순히 "루프")뿐만 아니라 스템 유사 ("비대합") 영역으로서 묘사된 영역을 포함하였고, 그리고 각각은 이차 Grik2 mRNA 구조 내에 염기 대합의 낮은 확률을 갖는다. 하나 이상의 종 (최소한 인간, 그리고 일부 경우에 적어도 하나의 추가 종 Grik2 전사체, 예컨대 생쥐 또는 원숭이)에서 낮은 염기 대합 확률 및/또는 높은 위치 엔트로피를 가질 것으로 예측된 영역이 우선적으로 처리되었다. 실제로, 예측된 이차 Grik2 mRNA 구조 (표 4 및 도 1b)의 많은 루프 및 스템 유사 비대합 영역은 RNAup 또는 동등한 계산에 의해 결정될 때, 다양한 siRNA 및 miRNA 가이드와의 대합 배열에서 호의적인 낮은 에너지 요구량, 예를 들면 10, 그리고 심지어 7.5 kcal/mol보다 작은 표적 개방 에너지를 나타내는 호의적인 영역을 내포한다 (참조: 실시예 1B, 표 12 및 표 13).
따라서, 본원 발명의 ASO 작용제 (예를 들면, 서열 번호: 1-108 중에서 하나)에 혼성화될 때 Grik2의 발현을 감소시킬 수 있는, Grik2 mRNA의 이차 구조 일부 또는 영역, 예를 들면, 루프 및 비대합 영역 내에 Grik2 표적 핵산이 확인되었고, 그리고 본원 발명의 구체예이다. Grik2 mRNA 내에 예시적인 이차 구조 영역에 대해 표 4 및 도 1b를 참조한다.
따라서, 개시된 ASO 작용제는 Grik2 mRNA 내에 이차 구조 (예를 들면, 루프 또는 비대합된 이차 구조)에 결합할 수 있다. 예를 들면, ASO 작용제는 Grik2 mRNA의 이차 구조 내에 루프 영역, 예컨대 예를 들면, 서열 번호: 115의 뉴클레오티드 위치 494-524 또는 서열 번호: 116의 위치 201-231에 위치된 루프 1 영역에 결합할 수 있다. 루프 1 영역은 서열 번호: 145의 핵산 서열을 가질 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 145의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다. 따라서, 개시된 ASO 작용제는 Grik2 mRNA의 루프 1 영역의 적어도 일부 내에 결합할 수 있다. 예를 들면, 서열 번호: 115 또는 116의 루프 1 영역 (서열 번호: 145)의 일부 또는 영역 내에 핵산 서열을 표적으로 하는 Grik2 ASO 작용제는 siRNA G0 (서열 번호: 1) 또는 siRNA G0 (서열 번호: 1)의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
다른 사례에서, ASO 작용제는 서열 번호: 115의 뉴클레오티드 위치 1098-1124 또는 서열 번호: 116의 위치 805-831에 위치된 루프 2 영역에 결합할 수 있다. 루프 2 영역은 서열 번호: 146의 핵산 서열을 가질 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 146의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다. 따라서, 개시된 ASO 작용제는 Grik2 mRNA의 루프 2 영역의 적어도 일부 내에 결합할 수 있다. 예를 들면, 서열 번호: 115 또는 116의 루프 2 영역 (서열 번호: 146)의 일부 또는 영역 내에 핵산 서열을 표적으로 하는 Grik2 ASO 작용제는 siRNA TT (서열 번호: 4) 또는 siRNA TT (서열 번호: 4)의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
ASO 작용제는 또한, 서열 번호: 115의 뉴클레오티드 위치 1197-1237 또는 서열 번호: 116의 위치 904-944에 위치된 루프 3 영역에 결합하는 것일 수 있다. 루프 3 영역은 서열 번호: 147의 핵산 서열을 가질 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 147의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다. 따라서, 개시된 ASO 작용제는 Grik2 mRNA의 루프 3 영역의 적어도 일부 내에 결합할 수 있다. 예를 들면, 서열 번호: 115 또는 116의 루프 3 영역 (서열 번호: 147)의 일부 또는 영역 내에 핵산 서열을 표적으로 하는 Grik2 ASO 작용제는 siRNA G1 (서열 번호: 5) 또는 siRNA G2 (서열 번호: 6), 또는 siRNA G1 (서열 번호: 5) 또는 siRNA G2 (서열 번호: 6)의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
추가 실례에서, ASO 작용제는 서열 번호: 115의 뉴클레오티드 위치 1543-1569 또는 서열 번호: 116의 위치 1250-1276에 위치된 루프 4 영역에 결합할 수 있다. 루프 4 영역은 서열 번호: 148의 핵산 서열을 가질 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 148의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다. 따라서, 개시된 ASO 작용제는 Grik2 mRNA의 루프 4 영역의 적어도 일부 내에 결합할 수 있다.
ASO 작용제는 또한, 서열 번호: 115의 뉴클레오티드 위치 1667-1731 또는 서열 번호: 116의 위치 1374-1438에 위치된 루프 5 영역에 결합하는 것일 수 있다. 루프 5 영역은 서열 번호: 149의 핵산 서열을 가질 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 149의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다. 따라서, 개시된 ASO 작용제는 Grik2 mRNA의 루프 5 영역의 적어도 일부 내에 결합할 수 있다. 예를 들면, 서열 번호: 115 또는 116의 루프 5 영역 (서열 번호: 149)의 일부 또는 영역 내에 핵산 서열을 표적으로 하는 Grik2 ASO 작용제는 siRNA GD (서열 번호: 7) 또는 siRNA MU (서열 번호: 96), 또는 siRNA GD (서열 번호: 7) 또는 siRNA MU (서열 번호: 96)의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
추가 실례에서, ASO 작용제는 서열 번호: 115의 뉴클레오티드 위치 1767-1830 또는 서열 번호: 116의 위치 1474-1537에 위치된 루프 6 영역에 결합하는 것일 수 있다. 루프 6 영역은 서열 번호: 150의 핵산 서열을 가질 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 150의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다. 따라서, 개시된 ASO 작용제는 Grik2 mRNA의 루프 6 영역의 적어도 일부 내에 결합할 수 있다. 예를 들면, 서열 번호: 115 또는 116의 루프 6 영역 (서열 번호: 150)의 일부 또는 영역 내에 핵산 서열을 표적으로 하는 Grik2 ASO 작용제는 siRNA G3 (서열 번호: 8), siRNA MS (서열 번호: 99) 또는 siRNA MT (서열 번호: 98), 또는 siRNA G3 (서열 번호: 8), siRNA MS (서열 번호: 99) 또는 siRNA MT (서열 번호: 98)의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
ASO 작용제는 또한, 서열 번호: 115의 뉴클레오티드 위치 2693-2716 또는 서열 번호: 116의 위치 2400-2423에 위치된 루프 7 영역에 결합하는 것일 수 있다. 루프 7 영역은 서열 번호: 151의 핵산 서열을 가질 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 151의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이다. 따라서, 개시된 ASO 작용제는 Grik2 mRNA의 루프 7 영역의 적어도 일부 내에 결합할 수 있다.
ASO 작용제는 또한, 서열 번호: 115의 뉴클레오티드 위치 2916-2955 또는 서열 번호: 116의 위치 2623-2662에 위치된 루프 8 영역에 결합하는 것일 수 있다. 루프 8 영역은 서열 번호: 152의 핵산 서열을 가질 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 152의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다. 따라서, 개시된 ASO 작용제는 Grik2 mRNA의 루프 8 영역의 적어도 일부 내에 결합할 수 있다. 예를 들면, 서열 번호: 115 또는 116의 루프 8 영역 (서열 번호: 152)의 일부 또는 영역 내에 핵산 서열을 표적으로 하는 Grik2 ASO 작용제는 siRNA G4 (서열 번호: 9) 또는 siRNA G4 (서열 번호: 9)의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
추가적으로, ASO 작용제는 서열 번호: 115의 뉴클레오티드 위치 3065-3091에 위치된 루프 9 영역에 결합하는 것일 수 있다. 루프 9 영역은 서열 번호: 153의 핵산 서열을 가질 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 153의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다. 따라서, 개시된 ASO 작용제는 Grik2 mRNA의 루프 9 영역의 적어도 일부 내에 결합할 수 있다. 예를 들면, 서열 번호: 115 또는 116의 루프 9 영역 (서열 번호: 153)의 일부 또는 영역 내에 핵산 서열을 표적으로 하는 Grik2 ASO 작용제는 siRNA YA (서열 번호: 63) 또는 siRNA YA (서열 번호: 63)의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
게다가, ASO 작용제는 서열 번호: 115의 뉴클레오티드 위치 3141-3163에 위치된 루프 10 영역에 결합하는 것일 수 있다. 루프 10 영역은 서열 번호: 154의 핵산 서열을 가질 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 154의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다. 따라서, 개시된 ASO 작용제는 Grik2 mRNA의 루프 10 영역의 적어도 일부 내에 결합할 수 있다.
추가 실례에서, ASO 작용제는 서열 번호: 115의 뉴클레오티드 위치 3382-3413에 위치된 루프 11 영역에 결합하는 것일 수 있다. 루프 11 영역은 서열 번호: 155의 핵산 서열을 가질 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 155의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다. 따라서, 개시된 ASO 작용제는 Grik2 mRNA의 루프 11 영역의 적어도 일부 내에 결합할 수 있다. 예를 들면, 서열 번호: 115 또는 116의 루프 11 영역 (서열 번호: 155)의 일부 또는 영역 내에 핵산 서열을 표적으로 하는 Grik2 ASO 작용제는 siRNA TS (서열 번호: 10) 또는 siRNA TS (서열 번호: 10)의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
ASO 작용제는 또한, 서열 번호: 115의 뉴클레오티드 위치 3788-3856에 위치된 루프 12 영역에 결합하는 것일 수 있다. 루프 12 영역은 서열 번호: 156의 핵산 서열을 가질 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 156의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다. 따라서, 개시된 ASO 작용제는 Grik2 mRNA의 루프 12 영역의 적어도 일부 내에 결합할 수 있다. 예를 들면, 서열 번호: 115 또는 116의 루프 2 영역 (서열 번호: 156)의 일부 또는 영역 내에 핵산 서열을 표적으로 하는 Grik2 ASO 작용제는 siRNA TR (서열 번호: 11) 또는 siRNA TR (서열 번호: 11)의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
ASO 작용제는 서열 번호: 115의 뉴클레오티드 위치 4550-4592에 위치된 루프 13 영역에 결합하는 것일 수 있다. 루프 13 영역은 서열 번호: 157의 핵산 서열을 가질 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 157의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다. 따라서, 개시된 ASO 작용제는 Grik2 mRNA의 루프 13 영역의 적어도 일부 내에 결합할 수 있다.
추가 실례에서, ASO 작용제는 서열 번호: 115의 뉴클레오티드 위치 4363-4386에 위치된 루프 14 영역에 결합하는 것일 수 있다. 루프 14 영역은 서열 번호: 158의 핵산 서열을 가질 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 158의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다. 따라서, 개시된 ASO 작용제는 Grik2 mRNA의 루프 14 영역의 적어도 일부 내에 결합할 수 있다.
대안으로, 개시된 ASO 작용제는 Grik2 mRNA의 이차 구조 내에 비대합 영역, 예컨대 예를 들면, 서열 번호: 115의 뉴클레오티드 위치 2209-2287 또는 서열 번호: 116의 위치 1916-1994에 위치된 비대합 영역 1에 결합하는 것일 수 있다. 비대합 영역 1은 서열 번호: 159의 핵산 서열을 가질 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 159의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다. 따라서, 본원 발명의 ASO 작용제는 Grik2 mRNA의 비대합 영역 1의 적어도 일부 내에 결합할 수 있다. 예를 들면, 서열 번호: 115 또는 116의 비대합 영역 1 (서열 번호: 159)의 일부 또는 영역 내에 핵산 서열을 표적으로 하는 Grik2 ASO 작용제는 siRNA TP (서열 번호: 13), siRNA TO (서열 번호: 14), siRNA MR (서열 번호: 72) 또는 siRNA MQ (서열 번호: 73), 또는 siRNA TP (서열 번호: 13), siRNA TO (서열 번호: 14), siRNA MR (서열 번호: 72) 또는 siRNA MQ (서열 번호: 73)의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
추가 실례에서, ASO 작용제는 서열 번호: 115의 뉴클레오티드 위치 2355-2391 또는 서열 번호: 116의 위치 2062-2098에 위치된 비대합 영역 2에 결합하는 것일 수 있다. 비대합 영역 2는 서열 번호: 160의 핵산 서열을 가질 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 160의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다. 따라서, 개시된 ASO 작용제는 Grik2 mRNA의 비대합 영역 2의 적어도 일부 내에 결합할 수 있다.
추가 실례로서, ASO 작용제는 서열 번호: 115의 뉴클레오티드 위치 3324-3368에 위치된 비대합 영역 3에 결합하는 것일 수 있다. 비대합 영역 3은 서열 번호: 161의 핵산 서열을 가질 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 161의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다. 따라서, 본원 발명의 ASO 작용제는 Grik2 mRNA의 비대합 영역 3의 적어도 일부 내에 결합할 수 있다. 예를 들면, 서열 번호: 115 또는 116의 비대합 영역 3 (서열 번호: 161)의 일부 또는 영역 내에 핵산 서열을 표적으로 하는 Grik2 ASO 작용제는 siRNA G5 (서열 번호: 15) 또는 siRNA G5 (서열 번호: 15)의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
본원 발명의 ASO 작용제는 또한, 서열 번호: 115의 뉴클레오티드 위치 3587-3639에 위치된 비대합 영역 4에 결합하는 것일 수 있다. 비대합 영역 4는 서열 번호: 162의 핵산 서열을 가질 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 162의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다. ASO 작용제는 Grik2 mRNA의 비대합 영역 4의 적어도 일부 내에 결합할 수 있다. 예를 들면, 서열 번호: 115 또는 116의 비대합 영역 4 (서열 번호: 162)의 일부 또는 영역 내에 핵산 서열을 표적으로 하는 Grik2 ASO 작용제는 siRNA TN (서열 번호: 16) 또는 siRNA TN (서열 번호: 16)의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
게다가, ASO 작용제는 서열 번호: 115의 뉴클레오티드 위치 3686-3713에 위치된 비대합 영역 5에 결합하는 것일 수 있다. 비대합 영역 5는 서열 번호: 163의 핵산 서열을 가질 수 있거나, 또는 표 4에서 도시된 바와 같은, 서열 번호: 163의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다. ASO 작용제는 Grik2 mRNA의 비대합 영역 5의 적어도 일부 내에 결합할 수 있다. 예를 들면, 서열 번호: 115 또는 116의 비대합 영역 5 (서열 번호: 163)의 일부 또는 영역 내에 핵산 서열을 표적으로 하는 Grik2 ASO 작용제는 siRNA TM (서열 번호: 17) 또는 siRNA TM (서열 번호: 17)의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다.
표 4: 표적
Grik2
mRNA 서열을 인코딩하는 cDNA 서열
본원 발명의 ASO 작용제는 또한, 서열 번호: 582-681 (표 2 참조)에서 선택되는 뉴클레오티드 서열에 의해 인코딩된 Grik2 mRNA (예를 들면, 서열 번호: 115)의 영역 중에서 하나에 또는 서열 번호: 164-581에서 설명된 뉴클레오티드 서열에 의해 인코딩된 Grik2 mRNA의 영역 중에서 하나에 완전한 또는 실제적인 상보성으로 결합할 수 있다. 예를 들면, 본원 발명의 ASO 작용제는 서열 번호: 582-681 또는 서열 번호: 582-681 중에서 하나의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에서 선택되는 Grik2 mRNA (예를 들면, 서열 번호: 115)의 영역 중에서 하나에 결합하는 것일 수 있다. 다른 실례에서, 본원 발명의 ASO 작용제는 서열 번호: 582-681 또는 서열 번호: 582-681 중에서 하나의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에서 선택되는 Grik2 mRNA (예를 들면, 서열 번호: 115)의 영역 중에서 하나에 결합하는 것일 수 있다. 다른 실례에서, 본원 발명의 ASO 작용제는 서열 번호: 582-681 또는 서열 번호: 582-681 중에서 하나의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에서 선택되는 Grik2 mRNA (예를 들면, 서열 번호: 115)의 영역 중에서 하나에 결합하는 것일 수 있다. 다른 실례에서, 본원 발명의 ASO 작용제는 서열 번호: 582-681에서 선택되는 Grik2 mRNA (예를 들면, 서열 번호: 115)의 영역 중에서 하나에 결합하는 것일 수 있다. 다른 실례에서, 본원 발명의 ASO 작용제는 서열 번호: 164-581 또는 서열 번호: 164-581 중에서 하나의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에서 선택되는 Grik2 mRNA (예를 들면, 서열 번호: 115)의 영역 중에서 하나에 결합하는 것일 수 있다. 다른 실례에서, 본원 발명의 ASO 작용제는 서열 번호: 164-581 또는 서열 번호: 164-581 중에서 하나의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에서 선택되는 Grik2 mRNA (예를 들면, 서열 번호: 115)의 영역 중에서 하나에 결합하는 것일 수 있다. 다른 실례에서, 본원 발명의 ASO 작용제는 서열 번호: 164-581 또는 서열 번호: 164-581 중에서 하나의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에서 선택되는 Grik2 mRNA (예를 들면, 서열 번호: 115)의 영역 중에서 하나에 결합하는 것일 수 있다. 다른 실례에서, 본원 발명의 ASO 작용제는 서열 번호: 164-581에서 선택되는 Grik2 mRNA (예를 들면, 서열 번호: 115)의 영역 중에서 하나에 결합하는 것일 수 있다.
변형된 올리고뉴클레오티드
본원에서 개시된 ASO 작용제는 자연 발생 및/또는 변형된 뉴클레오티드를 내포할 수 있다. 올리고뉴클레오티드는 생체내에서 안정성 및/또는 치료 효율을 증가시키기 위해 변형될 수 있다, 특히 화학적으로 변형될 수 있다. 본원 발명의 ASO 작용제의 효능을 향상시킬 변형, 예컨대 안정화 변형 및/또는 표적화된 전사체의 분해를 방지하기 위해 RNA분해효소 H 활성화를 감소시키는 변형은 당해 분야에서 널리 알려져 있다 (참조: 예를 들면, Bennett and Swayze, Annu. Rev. Pharmacol. Toxicol. 50:259-293, 2010; 및 Juliano, Nucleic Acids Res. 19;44(14):6518-48, 2016). 특히, 본원 발명의 맥락에서 이용되는 올리고뉴클레오티드는 변형된 뉴클레오티드를 포함할 수 있다. 화학적 변형은 3가지 상이한 부위에서: (i) 인산염 기에서, (ii) 당 모이어티 상에서, 및/또는 (iii) 올리고뉴클레오티드의 전체 백본 구조 상에서 일어날 수 있다. 전형적으로, 화학적 변형은 백본 변형, 헤테로환 변형, 당 변형, 그리고 접합 전략을 포함한다.
예를 들면 올리고뉴클레오티드는 올리고데옥시리보뉴클레오티드, 올리고리보뉴클레오티드, 작은 조절 RNA (sRNA), U7- 또는 U1 매개 ASO, 또는 이들의 접합체 산물 예컨대 펩티드-접합된 또는 나노입자-복합화된 ASO, 백본 변형에 의해 화학적으로 변형된 올리고뉴클레오티드 예컨대 모르폴리노, 포스포로디아미데이트 모르폴리노 소중합체 (포스포로디아미데이트 모르폴리노, PMO), 펩티드 핵산 (PNA), 포스포로티오에이트 (PS) 올리고뉴클레오티드, 입체화학적으로 순수한 포스포로티오에이트 (PS) 올리고뉴클레오티드, 포스포라미데이트 변형된 올리고뉴클레오티드, 티오포스포라미데이트-변형된 올리고뉴클레오티드 및 메틸포스포네이트 변형된 올리고뉴클레오티드; 헤테로환 변형에 의해 화학적으로 변형된 올리고뉴클레오티드 예컨대 이중환 변형된 올리고뉴클레오티드, 이중환상 핵산 (BNA), 삼환상 변형된 올리고뉴클레오티드, 트리시클로-DNA-안티센스 올리고뉴클레오티드 (ASO), 핵염기 변형 예컨대 피리미딘 핵염기 상에서 5-메틸 치환, 5-치환된 피리미딘 유사체, 2-티오-티민 변형된 올리고뉴클레오티드 및 퓨린 변형된 올리고뉴클레오티드; 당 변형에 의해 화학적으로 변형된 올리고뉴클레오티드 예컨대 잠금 핵산 (LNA) 올리고뉴클레오티드, 2',4'-메틸렌옥시 가교된 핵산 (BNA), 에틸렌-가교된 핵산 (ENA), 제약된 에틸 (cEt) 올리고뉴클레오티드, 2'-변형된 RNA, 2'- 및 4'-변형된 올리고뉴클레오티드 예컨대 2'-O-Me RNA (2'-OMe), 2'-O-메톡시에틸 RNA (MOE), 2'-플루오로 RNA (FRNA) 및 4'-티오-변형된 DNA와 RNA; 접합 전략에 의해 화학적으로 변형된 올리고뉴클레오티드 예컨대 N-아세틸 갈락토사민 (GalNAc) 올리고뉴클레오티드 접합체 예컨대 5'-GalNAc 및 3'-GalNAc ASO 접합체, 지질 올리고뉴클레오티드 접합체, 세포 투과성 펩티드 (CPP) 올리고뉴클레오티드 접합체, 표적화된 올리고뉴클레오티드 접합체, 항체-올리고뉴클레오티드 접합체, 중합체-올리고뉴클레오티드 접합체 예컨대 페길화 및 표적화 리간드를 갖는 접합체; 그리고 예를 들면 Bennett and Swayze, 2010 (위와 같음); Wan and Seth, J Med Chem. 59(21):9645-9667, 20116); Juliano, 2016 (위와 같음); Lundin et al., Hum Gene Ther. 26(8):475-485, 2015); 및 Prakash, Chem Biodivers. 8(9):1616-1641, 2011)에서 설명된 화학적 변형 및 접합 전략으로 구성된 군에서 선택될 수 있다. 실제로, 생체내에서 이용하기 위해, 올리고뉴클레오티드는 안정화될 수 있다. "안정화된" 올리고뉴클레오티드는 생체내 분해 (예를 들면, 엑소뉴클레아제 또는 엔도뉴클레아제를 통한)에 상대적으로 내성인 올리고뉴클레오티드를 지칭한다. 안정화는 길이 또는 이차 구조의 함수일 수 있다. 특히, 올리고뉴클레오티드 안정화는 인산염 백본 변형, 포스포디에스테르 변형, 포스포로티오에이트 (PS) 백본 변형, 포스포디에스테르 및 포스포로티오에이트 변형의 조합, 티오포스포라미데이트 변형, 2' 변형 (2'-O-Me, 2'-O-(2-메톡시에틸) (MOE) 변형 및 2'-플루오로 변형), 메틸포스포네이트, 메틸포스포로티오에이트, 포스포로디티오에이트, p-에톡시, 그리고 이들의 조합을 통해 달성될 수 있다.
예를 들면, 올리고뉴클레오티드는 포스포로티오에이트 유도체 (비가교 포스포릴 산소 원자의 황 원자로의 대체)로서 이용될 수 있는데, 이들은 뉴클레아제 절단에 대한 증가된 내성을 갖는다. 2'-메톡시에틸 (MOE) 변형 (예컨대 IONIS Pharmaceuticals에 의해 상업화된 변형된 백본) 또한 효과적이다. 부가적으로 또는 대안으로, 본원 발명의 올리고뉴클레오티드는 당의 2' 위치에서 치환, 특히 하기 화학적 변형: O-메틸 기 (2'-O-Me) 치환, 2-메톡시에틸 기 (2'-O-MOE) 치환, 플루오르 기 (2'-플루오로) 치환, 클로로 기 (2'-Cl) 치환, 브로모 기 (2'-Br) 치환, 시안 기 (2'-CN) 치환, 트리플루오로메틸 기 (2'-CF3) 치환, OCF3 기 (2'-OCF3) 치환, OCN 기 (2'-OCN) 치환, O-알킬 기 (2'-O-알킬) 치환, S-알킬 기 (2'-S-알킬) 치환, N-알킬 기 (2'-N-알킬) 치환, O-알케닐 기 (2'-O-알케닐) 치환, S-알케닐 기 (2'-S-알케닐) 치환, N-알케닐 기 (2'-N-알케닐) 치환, SOCH3 기 (2'-SOCH3) 치환, SO2CH3 기 (2'-SO2CH3) 치환, ONO2 기 (2'-ONO2) 치환, NO2 기 (2'-NO2) 치환, N3 기 (2'-N3) 치환 및/또는 NH2 기 (2'-NH2) 치환을 갖는 유도체인 완전히, 부분적으로 또는 조합으로, 변형된 뉴클레오티드를 포함할 수 있다. 부가적으로 또는 대안으로, 본원 발명의 올리고뉴클레오티드는, 리보오스 모이어티가 잠금 핵산 (LNA)을 생산하는 데 이용되고, 여기서 공유 가교가 리보오스의 2' 산소 및 4' 탄소 사이에 형성되어, 이것을 3'-엔도 입체형상에서 고정시키는 완전하게 또는 부분적으로 변형된 뉴클레오티드를 포함할 수 있다. 이들 분자는 생물학적 매질에서 극히 안정화되고, 예컨대 LNA가 단부 (갭머)에 위치되고 상보성 RNA 및 DNA와 단단한 하이브리드를 형성할 때 RNA분해효소 H를 활성화할 수 있다.
본원 발명의 맥락에서 이용되는 올리고뉴클레오티드는 LNA, 2'-OMe 유사체, 2'-O-Met, 2'-O-(2-메톡시에틸) (MOE) 소중합체, 2'-포스포로티오에이트 유사체, 2'-플루오로 유사체, 2'-Cl 유사체, 2'-Br 유사체, 2'-CN 유사체, 2'-CF3 유사체, 2'-OCF3 유사체, 2'-OCN 유사체, 2'-O-알킬 유사체, 2'-S-알킬 유사체, 2'-N-알킬 유사체, 2'-O-알케닐 유사체, 2'-S-알케닐 유사체, 2'-N-알케닐 유사체, 2'-SOCH3 유사체, 2'-SO2CH3 유사체, 2'-ONO2 유사체, 2'-NO2 유사체, 2'-N3 유사체, 2'-NH2 유사체, 트리시클로 (tc)-DNA, U7 짧은 핵 (sn) RNA, 트리시클로-DNA-올리고안티센스 분자, 그리고 이들의 조합으로 구성된 군에서 선택되는 변형된 뉴클레오티드를 포함할 수 있다 (2009년 4월 10일 자 제출된, U.S. 특허가출원 일련 번호 61/212,384: Tricyclo-DNA Antisense Oligonucleotides, Compositions and Methods for the Treatment of Disease, 이것의 전체 내용은 본원에서 참조로서 편입된다).
특히, 본원 발명에 따른 올리고뉴클레오티드는 LNA 올리고뉴클레오티드일 수 있다. 용어 "LNA" (잠금 핵산) (또는 "LNA 올리고뉴클레오티드")는 LNA 뉴클레오티드 및 LNA 유사체 뉴클레오티드로서 또한 지칭되는 하나 이상의 이중환상, 삼중환상 또는 다중환상 뉴클레오시드 유사체를 내포하는 올리고뉴클레오티드를 지칭한다. LNA 올리고뉴클레오티드, LNA 뉴클레오티드 및 LNA 유사체 뉴클레오티드는 국제 공개 번호 WO 99/14226 및 후속 출원; 국제 공개 번호 WO 00/56746, WO 00/56748, WO 00/66604, WO 01/25248, WO 02/28875, WO 02/094250, WO 03/006475; U.S. 특허 번호 6,043,060, 6268490, 6770748, 6639051, 그리고 U.S. 공개 번호 2002/0125241, 2003/0105309, 2003/0125241, 2002/0147332, 2004/0244840 및 2005/0203042에서 전반적으로 설명되는데, 이들 모두 본원에서 참조로서 편입된다. LNA 올리고뉴클레오티드 및 LNA 유사체 올리고뉴클레오티드는 예를 들면, Proligo LLC, 6200 Lookout Road, Boulder, CO 80301 USA로부터 상업적으로 가용하다.
본원 발명의 올리고뉴클레오티드의 다른 형태는 렌티바이러스 또는 아데노 관련 바이러스에 기초되지만 이들에 한정되지 않는 바이러스 전달 방법과 조합으로, 작은 핵 RNA 분자 예컨대 U1 또는 U7에 연결된 올리고뉴클레오티드 서열이다 (Denti, MA, et al, 2008; Goyenvalle, A, et al, 2004).
본원 발명의 올리고뉴클레오티드의 다른 형태는 펩티드 핵산 (PNA)이다. 펩티드 핵산에서, 올리고뉴클레오티드의 데옥시리보오스 백본이 당보다 펩티드와 더 유사한 백본으로 대체된다. 각 아단위, 또는 단량체는 이러한 백본에 부착된 자연 발생 또는 비-자연 발생 염기를 갖는다. 이와 같은 한 가지 백본은 아미드 결합을 통해 연결된 N-(2-아미노에틸)글리신의 반복 단위로 작제된다. 데옥시리보오스 백본으로부터 라디칼 이탈로 인해, 이들 화합물은 펩티드 핵산 (PNA)으로 명명되었다 (Dueholm et al., New J. Chem., 1997, 21, 19-31). PNA는 DNA와 RNA 둘 모두에 결합하여 PNA/DNA 또는 PNA/RNA 이중나선을 형성한다. 그 결과로 생긴 PNA/DNA 또는 PNA/RNA 이중나선은 Tm에 의해 결정될 때, 상응하는 DNA/DNA, DNA/RNA 또는 RNA/RNA 이중나선보다 큰 친화성으로 결합된다. 이러한 높은 열 안정성은 PNA에서 중성 백본에 기인한 전하 반발의 결여에 기인할지도 모른다. PNA의 중성 백본은 또한, PNA/DNA(RNA) 이중나선의 Tm이 염 농도와 실질적으로 무관하도록 야기한다. 따라서, PNA/DNA(RNA) 이중나선 상호작용은 이온 강도에 고도로 의존하는 DNA/DNA, DNA/RNA 또는 RNA/RNA 이중나선 상호작용에 비하여 추가 이점을 제공한다. 호모피리미딘 PNA는 상보성 DNA 또는 RNA에 역평행 배향으로 결합하여, 높은 열 안정성의 (PNA)2/DNA(RNA) 트리플렉스를 형성하는 것으로 밝혀졌다 (참조: 예를 들면, Egholm, et al., Science, 1991, 254, 1497; Egholm, et al., J. Am. Chem. Soc., 1992, 114, 1895; Egholm, et al., J. Am. Chem. Soc., 1992, 114, 9677). 증가된 친화성에 더하여, PNA는 DNA 또는 RNA에 증가된 특이성으로 결합하는 것으로 또한 밝혀졌다. PNA/DNA 이중나선 부정합이 DNA/DNA 이중나선에 상대적으로 용융될 때, Tm에서 8 내지 20℃ 하락이 목격된다. Tm에서 이러한 규모의 하락은 부정합이 존재하는 상응하는 DNA/DNA 이중나선에서는 목격되지 않는다. DNA 또는 RNA 가닥에 PNA 가닥의 결합은 2가지 배향 중 하나에서 일어날 수 있다. 배향은 PNA의 카르복실 단부가 DNA 또는 RNA의 5' 단부를 향해 지향되고 PNA의 아미노 단부가 DNA 또는 RNA의 3' 단부를 향해 지향되도록, DNA 또는 RNA 가닥이 5'에서 3' 방향으로 상보성 PNA 가닥에 결합할 때, 역평행하는 것으로 일컬어진다. 평행 배향에서 PNA의 카르복실 단부 및 아미노 단부는 DNA 또는 RNA의 5'-3' 방향으로 정반대로 위치한다. 올리고뉴클레오티드와 비교하여 PNA의 추가 이점은 그들의 폴리아미드 백본 (적합한 핵염기 또는 다른 측쇄 기가 거기에 부착됨)이 뉴클레아제 또는 프로테아제 중 어느 것에 의해서도 인식되지 않고 개열되지 않는다는 것이다. 결과적으로, PNA는 핵산 및 펩티드와 달리 효소에 의한 분해에 내성이다. WO 92/20702는 상응하는 DNA보다 상보성 DNA와 RNA에 더 단단하게 결합하는 펩티드 핵산 (PNA) 화합물을 설명한다. PNA는 상보성 DNA에 대해 강한 결합 친화성과 특이성을 보여주었다 (Egholm, M., et al., Chem. Soc., Chem. Commun., 1993, 800; Egholm, M., et.al., Nature, 1993, 365, 566; 및 Nielsen, P., et.al. Nucl. Acids Res., 1993, 21, 197). 게다가, PNA는 세포 추출물에서 뉴클레아제 내성 및 안정성을 보여준다 (Demidov, V. V., et al., Biochem. Pharmacol., 1994, 48, 1309-1313). PNA의 변형은 확장된 백본 (Hyrup, B., et.al. Chem. Soc., Chem. Commun., 1993, 518), 백본 및 핵염기 사이에 확장된 링커, 아미다 결합의 반전 (Lagriffoul, P. H., et.al., Biomed. Chem. Lett., 1994, 4, 1081), 그리고 알라닌에 기초된 키랄 백본의 이용 (Dueholm, K. L, et.al., Biomed. Chem. Lett., 1994, 4, 1077)을 포함한다. 펩티드 핵산은 U.S. 특허 번호 5,539,082 및 U.S. 특허 번호 5,539,083에서 설명된다. 펩티드 핵산은 U.S. 특허 번호 5,766,855에서 더욱 설명된다.
본원 발명의 올리고뉴클레오티드 (예를 들면, ASO 작용제)는 당해 분야에서 널리 알려진 전통적인 방법에 의해 획득될 수 있다. 예를 들면, 본원 발명의 올리고뉴클레오티드는 당해 분야에서 널리 알려진 다수의 절차 중 임의의 것을 이용하는 데노보 합성될 수 있다. 예를 들면, b-시아노에틸 포스포라미디트 방법 (Beaucage et al., 1981); 뉴클레오시드 H-포스포네이트 방법 (Garegg et al., 1986; Froehler et al., 1986, Garegg et al., 1986, Gaffney et al., 1988). 이들 화학은 시장에서 구입 가능한 다양한 자동화 핵산 합성장치에 의해 수행될 수 있다. 이들 핵산은 합성 핵산으로서 지칭될 수 있다. 대안으로, 올리고뉴클레오티드는 플라스미드에서 대규모로 생산될 수 있다 (참조: Sambrook, et al., 1989). 올리고뉴클레오티드는 공지된 기술, 예컨대 제한 효소, 엑소뉴클레아제 또는 엔도뉴클레아제를 이용하는 기술을 이용하여, 기존의 핵산 서열로부터 제조될 수 있다. 이러한 방식으로 제조된 올리고뉴클레오티드는 단리된 핵산으로서 지칭될 수 있다.
올리고뉴클레오티드의 전달 및 효능을 증강하기 위한 접근방식 및 변형 예컨대 올리고뉴클레오티드를 표적화 작용제 예컨대 탄수화물, 펩티드, 항체, 앱타머, 지질 또는 소형 분자 및 올리고뉴클레오티드 전달을 향상시키는 소형 분자에 연결함에 의한, 올리고뉴클레오티드, 지질 및 중합체 기반 나노입자 또는 나노운반체, 리간드-올리고뉴클레오티드 접합체의 화학적 변형은 당해 분야에서 널리 알려져 있다, 예컨대 Juliano (2016; 위와 같음)에서 설명된다. 친유성 접합체 및 지질 접합체는 지방산-올리고뉴클레오티드 접합체; 스테롤-올리고뉴클레오티드 접합체 및 비타민-올리고뉴클레오티드 접합체를 포함한다.
본원 발명의 올리고뉴클레오티드는 또한, WO 2014/195432에서 설명된 바와 같이, 2 내지 30개의 탄소 원자, 특히 5 내지 20개의 탄소 원자, 더욱 특히 10 내지 18개의 탄소 원자를 포함하는 적어도 3개의 포화되거나 또는 불포화된, 특히 포화된, 선형 또는 분지형, 특히 선형 탄화수소 사슬을 포함하는 모이어티에 의한 3' 또는 5' 단부에서 치환에 의해 변형될 수 있다.
본원 발명의 올리고뉴클레오티드는 적어도 하나의 케탈 기능기를 포함하는 모이어티에 의한 3' 또는 5' 단부에서 치환에 의해 변형될 수 있는데, 여기서 상기 케탈 기능기의 케탈 탄소는 WO 2014/195430에서 설명된 바와 같이, 1 내지 22개의 탄소 원자, 특히 6 내지 20개의 탄소 원자, 특히 10 내지 19개의 탄소 원자, 더더욱 특히 12 내지 18개의 탄소 원자를 포함하는 2개의 포화되거나 또는 불포화된, 특히 포화된, 선형 또는 분지형, 특히 선형 탄화수소 사슬을 보유한다.
추가적으로, 본원 발명의 올리고뉴클레오티드는 두 번째 분자에 접합될 수 있다. 전형적으로, 두 번째 분자는 앱타머, 항체, 또는 폴리펩티드로 구성된 군에서 선택될 수 있다. 예를 들면, 본원 발명의 올리고뉴클레오티드는 세포 투과성 펩티드에 접합될 수 있다. 세포 투과성 펩티드는 당해 분야에서 널리 알려져 있고 예를 들면 TAT 펩티드를 포함한다 (참조: 예를 들면, Bechara and Sagan, FEBS Lett. 587(12):1693-1702, 2013).
올리고뉴클레오티드 작용제의 포유류 세포로의 전달
본원 발명의 올리고뉴클레오티드는 또한, 당해 분야에서 공지된 중합성, 생물분해성 마이크로입자, 또는 마이크로캡슐 전달 장치를 포함하는 다양한 막성 분자 조립체 전달 방법을 이용하여 전달될 수 있다. 예를 들면, 콜로이드 분산 시스템이 본원에서 설명된 올리고뉴클레오티드 작용제의 표적화된 전달에 이용될 수 있다. 콜로이드 분산 시스템은 거대분자 복합체, 나노캡슐, 마이크로스피어, 비드, 그리고 수중유 유제, 미셀, 혼합된 미셀 및 리포솜을 비롯한 지질 기반 시스템을 포함한다. 리포솜은 시험관내와 생체내 전달 수송체로서 유용한 인공 막 소포이다. 크기에서 0.2-4.0 μm 범위인 큰 단층판 소포 (LUV)는 큰 거대분자를 내포하는 수성 완충액의 실제적인 백분율을 캡슐화할 수 있는 것으로 밝혀졌다. 리포솜은 활성 성분의 작용 부위로의 이전 및 전달에 유용하다. 리포솜 막이 생물학적 막과 구조적으로 유사하기 때문에, 리포솜이 조직에 적용될 때, 리포솜 이중층은 세포 막의 이중층과 융합한다. 리포솜 및 세포의 합병이 진행됨에 따라서, 올리고뉴클레오티드를 포함하는 내부 수성 내용물은 올리고뉴클레오티드가 표적 RNA에 특이적으로 결합할 수 있고 RNA분해효소 H 매개 유전자 침묵을 매개할 수 있는 세포 내로 전달된다. 일부 경우에, 리포솜은 또한, 예를 들면, 올리고뉴클레오티드를 특정 세포 유형으로 지향시키기 위해 특이적으로 표적화된다. 리포솜의 조성은 통상적으로 스테로이드, 특히 콜레스테롤과 조합된, 인지질의 조합이다, 통상적으로 된다. 다른 인지질 또는 다른 지질 역시 이용될 수 있다. 리포솜의 물리적 특징은 pH, 이온 강도, 그리고 이가 양이온의 존재에 좌우된다.
올리고뉴클레오티드를 내포하는 리포솜은 다양한 방법에 의해 제조될 수 있다. 한 가지 실례에서, 미셀이 지질 성분으로 형성되도록, 리포솜의 지질 성분이 세정제에서 용해된다. 예를 들면, 지질 성분은 양친매성 양이온성 지질 또는 지질 접합체일 수 있다. 세정제는 높은 임계적 미셀 농도를 가질 수 있고 비이온성일 수 있다. 예시적인 세정제는 담즙산염, CHAPS, 옥틸글루코시드, 데옥시콜레이트, 그리고 라우로일 사르코신을 포함한다. 올리고뉴클레오티드 제조물이 이후, 지질 성분을 포함하는 미셀에 첨가된다. 지질 상에서 양이온성 기가 올리고뉴클레오티드와 상호작용하고 올리고뉴클레오티드 주변에서 축합되어 리포솜을 형성한다. 축합 후, 세정제가 예를 들면, 투석에 의해 제거되어, 올리고뉴클레오티드의 리포솜 제조물이 산출된다.
필요하면, 축합을 보조하는 운반체 화합물이 예를 들면, 제어된 첨가에 의해, 축합 반응 동안 첨가될 수 있다. 예를 들면, 운반체 화합물은 핵산 (예를 들면, 스페르민 또는 스페르미딘) 이외의 중합체일 수 있다. pH 또한, 축합에 호의적이도록 조정될 수 있다.
전달 수송체의 구조 성분으로서 폴리뉴클레오티드/양이온성 지질 복합체를 통합하는 안정된 폴리뉴클레오티드 전달 수송체를 생산하기 위한 방법은 예를 들면, WO 96/37194에서 더욱 설명되는데, 이것의 전체 내용은 본원에서 참조로서 편입된다. 리포솜 형성은 Feigner, P. L. et al., (1987) Proc. Natl. Acad. Sci. USA 8:7413-7417; U.S. 특허 번호 4,897,355; U.S. 특허 번호 5,171,678; Bangham et al., (1965) M. Mol. Biol. 23:238; Olson et al., (1979) Biochim. Biophys. Acta 557:9; Szoka et al., (1978) Proc. Natl. Acad. Sci. 75: 4194; Mayhew et al., (1984) Biochim. Biophys. Acta 775:169; Kim et al., (1983) Biochim. Biophys. Acta 728:339; 및 Fukunaga et al., (1984) Endocrinol. 115:757에서 설명된 예시적인 방법의 하나 이상의 양상을 또한 포함할 수 있다. 전달 수송체로서 이용하기 위한 적합한 크기의 지질 응집체를 제조하기 위해 통상적으로 이용되는 기술은 초음파처리 및 동결 해동 플러스 압출을 포함한다 (참조: 예를 들면, Mayer et al., (1986) Biochim. Biophys. Acta 858:161. 일관되게 작고 (50 내지 200 nm) 상대적으로 균일한 응집체가 요망될 때, 미세유동화가 이용될 수 있다 (Mayhew et al., (1984) Biochim. Biophys. Acta 775:169. 이들 방법은 올리고뉴클레오티드 제조물을 리포솜 내로 포장하는 데 쉽게 적응된다.
리포솜은 2가지 광범위한 부류에 속한다. 양이온성 리포솜은 음으로 하전된 핵산 분자와 상호작용하여 안정된 복합체를 형성하는, 양으로 하전된 리포솜이다. 양으로 하전된 핵산/리포솜 복합체는 음으로 하전된 세포 표면에 결합하고 엔도솜에 내재화된다. 엔도솜 내에 산성 pH로 인해, 리포솜은 파열되어, 그들의 내용물이 세포 세포질 내로 방출된다 (Wang et al. (1987) Biochem. Biophys. Res. Commun., 147:980-985).
pH 민감성이거나 또는 음으로 하전된 리포솜은 핵산과 복합체를 이루기보다는 이들을 가둔다. 핵산 및 지질 둘 모두 유사하게 하전되기 때문에, 복합체 형성보다는 반발이 발생한다. 그럼에도 불구하고, 일부 핵산은 이들 리포솜의 수성 내부 내에 갇힌다. pH 민감성 리포솜이 티미딘 키나아제 유전자를 인코딩하는 핵산을 배양 중인 세포 단층에 전달하는 데 이용되었다. 외인성 유전자의 발현이 표적 세포에서 검출되었다 (Zhou et al. (1992) Journal of Controlled Release, 19:269-274).
리포솜 조성물의 한 가지 주요 유형은 자연적으로 유래된 포스파티딜콜린 이외의 인지질을 포함한다. 중성 리포솜 조성물은 예를 들면, 디미리스토일 포스파티딜콜린 (DMPC) 또는 디팔미토일 포스파티딜콜린 (DPPC)으로부터 형성될 수 있다. 음이온성 리포솜 조성물은 일반적으로 디미리스토일 포스파티딜글리세롤로부터 형성되는 반면, 음이온성 융해성 리포솜은 일차적으로 디올레오일 포스파티딜에탄올아민 (DOPE)으로부터 형성된다. 다른 유형의 리포솜 조성물은 포스파티딜콜린 (PC) 예컨대 예를 들면, 대두 PC 및 계란 PC로부터 형성된다. 다른 유형은 인지질 및/또는 포스파티딜콜린 및/또는 콜레스테롤의 혼합물로부터 형성된다.
시험관내와 생체내에서 리포솜을 세포 내로 도입하기 위한 다른 방법의 실례는 U.S. 특허 번호 5,283,185; U.S. 특허 번호 5,171,678; WO 94/00569; WO 93/24640; WO 91/16024; Feigner, (1994) J. Biol. Chem. 269:2550; Nabel, (1993) Proc. Natl. Acad. Sci. 90:11307; Nabel, (1992) Human Gene Ther. 3:649; Gershon, (1993) Biochem. 32:7143; 및 Strauss, (1992) EMBO J. 11:417을 포함한다.
리포솜은 또한, 하나 이상의 특수한 지질을 포함하는 입체적으로 안정화된 리포솜일 수 있는데, 이들 특수한 지질은 이런 특수한 지질을 결여하는 리포솜에 비하여 증강된 순환 수명을 야기한다. 입체적으로 안정화된 리포솜의 실례는 리포솜의 소포-형성 지질 부분의 일부가 (A) 하나 이상의 당지질, 예컨대 모노시알로강글리오시드 GM1을 포함하거나, 또는 (B) 하나 이상의 친수성 중합체, 예컨대 폴리에틸렌 글리콜 (PEG) 모이어티로 유도체화되는 것들이다. 임의의 특정 이론에 한정됨 없이, 당해 분야에서, 적어도, 강글리오시드, 스핑고미엘린, 또는 PEG 유도체화 지질을 내포하는 입체적으로 안정화된 리포솜의 경우에, 이들 입체적으로 안정화된 리포솜의 증강된 순환 반감기는 세망내피계 (RES)의 세포 내로 감소된 흡수에서 파생하는 것으로 생각된다 (Allen et al., (1987) FEBS Letters, 223:42; Wu et al., (1993) Cancer Research, 53:3765).
하나 이상의 당지질을 포함하는 다양한 리포솜이 당해 분야에서 알려져 있다. Papahadjopoulos et al. (Ann. N.Y. Acad. Sci., (1987), 507:64)은 리포솜의 혈액 반감기를 향상시키는, 모노시알로강글리오시드 GM1, 갈락토세레브로시드 황산염 및 포스파티딜이노시톨의 능력을 보고하였다. 이들 조사 결과는 Gabizon et al. (Proc. Natl. Acad. Sci. U.S.A., (1988), 85:6949)에 의해 자세히 설명되었다. U.S. 특허 번호 4,837,028 및 WO 88/04924 (둘 모두, Allen et al.)는 (1) 스핑고미엘린 및 (2) 강글리오시드 GM1 또는 갈락토세레브로시드 황산염 에스테르를 포함하는 리포솜을 개시한다. U.S. 특허 번호 5,543,152 (Webb et al.)는 스핑고미엘린을 포함하는 리포솜을 개시한다. 1,2-sn-디미리스토일포스파티딜콜린을 포함하는 리포솜은 WO 97/13499 (Lim et al)에서 개시된다.
본원 발명에 따라서, 양이온성 리포솜은 약물 전달 수송체로서 이용될 수 있다. 양이온성 리포솜은 세포막에 융합할 수 있는 이점을 소유한다. 비양이온성 리포솜은 비록 원형질막과 효율적으로 융합할 수 있는 것은 아니지만, 생체내에서 대식세포에 의해 흡수되고, 그리고 올리고뉴클레오티드를 대식세포에 전달하는 데 이용될 수 있다.
리포솜의 추가 이점은 다음을 포함한다: (i) 천연 인지질로부터 획득된 리포솜은 생체적합성이고 생물분해성이다; (ii) 리포솜은 넓은 범위의 수용성 및 지용성 약물을 통합할 수 있다; 그리고 (iii) 리포솜은 그들의 내부 구획 내에 캡슐화된 올리고뉴클레오티드를 대사 및 분해로부터 보호할 수 있다 (Rosoff, in "Pharmaceutical Dosage Forms," Lieberman, Rieger and Banker (Eds.), 1988, volume 1, p. 245). 리포솜 제제의 제조에서 중요한 고려 사항은 리포솜의 지질 표면 전하, 소포 크기 및 수성 용적이다.
양으로 하전된 합성 양이온성 지질인 N-[1-(2,3-디올레일옥시)프로필]-N,N,N-트리메틸암모늄 염화물 (DOTMA)은, 핵산과 자발적으로 상호작용하여 지질-핵산 복합체를 형성하는 작은 리포솜을 형성하는 데 이용될 수 있는데, 이들 복합체는 조직 배양 세포의 세포막의 음으로 하전된 지질과 융합하여, 올리고뉴클레오티드의 전달을 야기할 수 있다 (DOTMA 및 DNA와 함께 이의 이용에 관한 설명을 위해, 예를 들면, Feigner, P. L. et al., (1987) Proc. Natl. Acad. Sci. USA 8:7413-7417, 그리고 U.S. 특허 번호 4,897,355를 참조한다).
DOTMA 유사체인 1,2-비스(올레오일옥시)-3-(트리메틸암모니아)프로판 (DOTAP)은 인지질과 병용되어 DNA-복합화 소포를 형성할 수 있다. LIPOFECTINTM Bethesda Research Laboratories, Gaithersburg, Md.)는 음으로 하전된 폴리뉴클레오티드와 자발적으로 상호작용하여 복합체를 형성하는 양으로 하전된 DOTMA 리포솜을 포함하는 살아있는 조직 배양 세포 내로 고도로 음이온성 핵산의 전달을 위한 효과적인 작용제이다. 필요한 만큼의 양으로 하전된 리포솜이 이용될 때, 결과의 복합체 상에서 순전하 역시 양성이다. 이러한 방식으로 제조된 양으로 하전된 복합체는 음으로 하전된 세포 표면에 자발적으로 부착하고, 원형질막과 융합하고, 그리고 기능적 핵산을 예를 들면, 조직 배양 세포 내로 효율적으로 전달한다. 다른 상업적으로 가용한 양이온성 지질, 1,2-비스(올레오일옥시)-3,3-(트리메틸암모니아)프로판 ("DOTAP") (Boehringer Mannheim, Indianapolis, Ind.)은 올레오일 모이어티가 에테르 연쇄보다는 에스테르 연쇄에 의해 연결된다는 점에서 DOTMA와 상이하다.
다른 보고된 양이온성 지질 화합물은 예를 들면, 두 가지 유형의 지질 중에서 하나에 접합된 카르복시스페르민을 비롯하여, 다양한 모이어티에 접합된 것들을 포함하고, 그리고 5-카르복시스페르밀글리신 디옥타올레오일아미드 ("DOGS") (TRANSFECTAMTM, Promega, Madison, Wis.) 및 디팔미토일포스파티딜에탄올아민 5-카르복시스페르밀-아미드 ("DPPES")와 같은 화합물을 포함한다 (참조: 예를 들면, U.S. 특허 번호 5,171,678).
다른 양이온성 지질 접합체는 DOPE와 조합으로 리포솜으로 제형화된 콜레스테롤 ("DC-Chol")로 지질의 유도체화를 포함한다 (참조: Gao, X. and Huang, L., (1991) Biochim. Biophys. Res. Commun. 179:280). 폴리리신을 DOPE에 접합함으로써 만들어진 리포폴리리신은 혈청의 존재에서 형질감염에 효과적인 것으로 보고되었다 (Zhou, X. et al., (1991) Biochim. Biophys. Acta 1065:8). 일정한 세포주의 경우에, 접합된 양이온성 지질을 내포하는 이들 리포솜은 DOTMA 내포 조성물보다 더 낮은 독성을 나타내고 더 효율적인 형질감염을 제공하는 것으로 일컬어진다. 다른 상업적으로 가용한 양이온성 지질 제품은 DMRIE 및 DMRIE-HP (Vical, La Jolla, Calif.), 그리고 리포펙타민 (DOSPA) (Life Technology, Inc., Gaithersburg, Md.)을 포함한다. 올리고뉴클레오티드의 전달에 적합한 다른 양이온성 지질은 WO 98/39359 및 WO 96/37194에서 설명된다.
리포솜의 표적화 또한, 예를 들면, 기관 특이성, 세포 특이성 및 소기관 특이성에 근거하여 가능하고 당해 분야에서 알려져 있다. 리포솜 표적화된 전달 시스템의 경우에, 표적화 리간드를 리포솜 이중층과 안정된 연관 상태로 유지하기 위해, 지질 기가 리포솜의 지질 이중층 내로 통합될 수 있다. 다양한 연결기가 지질 사슬을 표적화 리간드에 연결하는 데 이용될 수 있다. 추가의 방법은 당해 분야에서 알려져 있고 예를 들면 U.S. 특허 출원 공개 번호 20060058255에서 설명되는데, 이의 연결기는 본원에서 참조로서 편입된다.
올리고뉴클레오티드, 예를 들면, 본원에서 설명된 ASO 작용제를 포함하는 리포솜은 고도로 변형 가능하도록 만들어질 수 있다. 이런 변형가능성은 리포솜이 리포솜의 평균 반지름보다 작은 포어를 침투할 수 있게 할 수 있다. 예를 들면, 트랜스퍼솜은 리포솜의 또 다른 유형이고, 그리고 약물 전달 수송체에 대한 매력적인 후보인 고도로 변형 가능한 지질 응집체이다. 트랜스퍼솜은 고도로 변형 가능하여 지질 방울보다 작은 포어를 쉽게 침투할 수 있는 지질 방울로서 설명될 수 있다. 트랜스퍼솜은 표면 가장자리 활성화제, 통상적으로 계면활성제를 표준 리포솜 조성물에 첨가함으로써 만들어질 수 있다. 올리고뉴클레오티드를 포함하는 트랜스퍼솜은 예를 들면, 올리고뉴클레오티드를 피부 내에 각질세포에 전달하기 위해 감염에 의해 피하 전달될 수 있다. 무손상 포유류 피부를 교차하기 위해, 지질 소포는 적절한 경피 구배의 영향하에, 각각 50 nm 미만의 직경을 갖는 일련의 미세한 포어를 통과해야 한다. 이에 더하여, 지질 특성으로 인해, 이들 트랜스퍼솜은 자가 최적화 (예를 들면, 피부 내에 포어의 모양에 적응성)될 수 있고, 자가 수복할 수 있고, 그리고 단편화 및 종종 자가 부하 없이 그들의 표적에 빈번하게 도달할 수 있다. 트랜스퍼솜은 혈청 알부민을 피부에 전달하는 데 이용되었다. 혈청 알부민의 트랜스퍼솜 매개 전달은 혈청 알부민을 내포하는 용액의 피하 주사만큼 효과적인 것으로 밝혀졌다.
본원 발명에 따른 다른 제제는 2008년 1월 2일 자 제출된 U.S. 가출원 일련 번호 61/018,616; 2008년 1월 2일 자 제출된 61/018,611; 2008년 3월 26일 자 제출된 61/039,748; 2008년 4월 22일 자 제출된 61/047,087 및 2008년 5월 8일 자 제출된 61/051,528에서 설명된다. 2007년 10월 3일 자 제출된 PCT 출원 번호 PCT/US2007/080331 역시 본원 발명에 따른 제제를 설명한다.
계면활성제는 유제 (마이크로유제 포함) 및 리포솜과 같은 제제에서 폭넓게 적용된다. 많은 상이한 유형의 계면활성제 (천연과 합성 둘 모두)의 특성을 분류하고 순위 매기는 가장 흔한 방식은 친수성/친유성 균형 (HLB)의 이용에 의한다. 친수성 기 ("머리"로서 또한 알려져 있음)의 성격은 제제에서 이용된 상이한 계면활성제를 분류하기 위한 가장 유용한 수단을 제공한다 (Rieger, in Pharmaceutical Dosage, Marcel Dekker, Inc., New York, N.Y., 1988, p. 285).
만약 계면활성제 분자가 이온화되지 않으면, 이것은 비이온성 계면활성제로서 분류된다. 비이온성 계면활성제는 약학 제품 및 미용 제품에서 폭넓게 적용되고 넓은 범위의 pH 값에 걸쳐 이용 가능하다. 일반적으로, 이들의 HLB 값은 그들의 구조에 따라서 2 내지 약 18의 범위 안에 있다. 비이온성 계면활성제는 비이온성 에스테르 예컨대 에틸렌 글리콜 에스테르, 프로필렌 글리콜 에스테르, 글리세릴 에스테르, 폴리글리세릴 에스테르, 소르비탄 에스테르, 수크로오스 에스테르, 그리고 에톡실화된 에스테르를 포함한다. 비이온성 알칸올아미드 및 에테르 예컨대 지방 알코올 에톡실레이트, 프로폭실화된 알코올, 그리고 에톡실화된/프로폭실화된 블록 중합체 또한 이러한 부류 내에 포함된다. 폴리옥시에틸렌 계면활성제는 비이온성 계면활성제 부류의 가장 대중적인 구성원이다.
만약 계면활성제 분자가 물에서 용해되거나 또는 분산될 때 음전하를 보유하면, 계면활성제는 음이온성으로서 분류된다. 음이온성 계면활성제는 카르복실산염 예컨대 비누, 아실 락틸레이트, 아미노산의 아실 아미드, 황산의 에스테르 예컨대 알킬 황산염 및 에톡실화된 알킬 황산염, 술폰산염 예컨대 알킬 벤젠 술폰산염, 아실 이세티오네이트, 아실 타우레이트 및 술포숙시네이트, 그리고 인산염을 포함한다. 음이온성 계면활성제 부류의 가장 중요한 구성원은 알킬 황산염 및 비누이다.
만약 계면활성제 분자가 물에서 용해되거나 또는 분산될 때 양전하를 보유하면, 계면활성제는 양이온성으로서 분류된다. 양이온성 계면활성제는 사차 암모늄 염 및 에톡실화된 아민을 포함한다. 사차 암모늄 염은 이러한 부류의 가장 많이 이용되는 구성원이다.
만약 계면활성제 분자가 양성 또는 음성 전하 중 어느 하나를 보유하는 능력을 갖는다면, 계면활성제는 양쪽성으로서 분류된다. 양쪽성 계면활성제는 아크릴산 유도체, 치환된 알킬아미드, N-알킬베타인, 그리고 인지질을 포함한다.
의약품, 제제 및 유제에서 계면활성제의 이용이 검토되었다 (Rieger, in Pharmaceutical Dosage Forms, Marcel Dekker, Inc., New York, N.Y., 1988, p. 285).
본원 발명의 방법에 이용하기 위한 올리고뉴클레오티드는 미셀 제제로서 또한 제공될 수 있다. 미셀은 분자의 모든 소수성 부분이 안쪽으로 지향되고 친수성 부분이 주변 수성 상과 접촉하도록, 양친매성 분자가 구형 구조로 배열되는 특정한 유형의 분자 조립체이다. 만약 주변 환경이 소수성이면, 정반대 배열이 존재한다.
지질 나노입자 기반 전달 방법
본원 발명의 올리고뉴클레오티드는 지질 제제, 예를 들면, 지질 나노입자 (LNP), 또는 다른 핵산-지질 입자 내에 완전히 캡슐화될 수 있다. LNP는 전신 적용에 극히 유용한데, 그 이유는 이들이 정맥내 주사 이후에 연장된 순환 수명을 나타내고 원위 부위 (예를 들면, 투여 부위로부터 물리적으로 분리된 부위)에서 축적되기 때문이다. LNP는 "pSPLP"를 포함하는데, 이들은 PCT 공개 번호 WO 00/03683에서 진술된 바와 같은 캡슐화된 축합제-핵산 복합체를 포함한다. 본원 발명의 입자는 전형적으로 약 50 nm 내지 약 150 nm, 더 전형적으로 약 60 nm 내지 약 130 nm, 더 전형적으로 약 70 nm 내지 약 110 nm, 가장 전형적으로 약 70 nm 내지 약 90 nm의 평균 직경을 갖고, 그리고 실제적으로 비독성이다. 이에 더하여, 핵산은 본원 발명의 핵산-지질 입자 내에 존재할 때, 수성 용액에서 뉴클레아제로 분해에 내성이다. 핵산-지질 입자 및 이들의 제조 방법은 예를 들면, U.S. 특허 번호 5,976,567; 5,981,501; 6,534,484; 6,586,410; 6,815,432; U.S. 공개 번호 2010/0324120 및 PCT 공개 번호 WO 96/40964에서 개시된다.
지질 대 약물 비율 (질량/질량 비율) (예를 들면, 지질 대 올리고뉴클레오티드 비율)은 약 1:1 내지 약 50:1, 약 1:1 내지 약 25:1, 약 3:1 내지 약 15:1, 약 4:1 내지 약 10:1, 약 5:1 내지 약 9:1, 또는 약 6:1 내지 약 9:1의 범위 안에 있을 수 있다. 상기 언급된 범위의 중간 범위 역시 본원 발명의 일부인 것으로 예기된다.
양이온성 지질의 무제한적 실례는 N,N-디올레일-N,N-디메틸암모늄 염화물 (DODAC), N,N-디스테아릴-N,N-디메틸암모늄 브롬화물 (DDAB), N--(I-(2,3-디올레오일옥시)프로필)-N,N,N-트리메틸암모늄 염화물 (DOTAP), N--(I-(2,3-디올레일옥시)프로필)-N,N,N-트리메틸암모늄 염화물 (DOTMA), N,N-디메틸-2,3-디올레일옥시)프로필아민 (DODMA), 1,2-디리놀레일옥시-N,N-디메틸아미노프로판 (DLinDMA), 1,2-디리놀레닐옥시-N,N-디메틸아미노프로판 (DLenDMA), 1,2-디리놀레일카르바모일옥시-3-디메틸아미노프로판 (DLin-C-DAP), 1,2-디리놀레일옥시-3-(디메틸아미노)아세톡시프로판 (DLin-DAC), 1,2-디리놀레일옥시-3-모르폴리노프로판 (DLin-MA), 1,2-디리놀레오일-3-디메틸아미노프로판 (DLinDAP), 1,2-디리놀레일티오-3-디메틸아미노프로판 (DLin-S-DMA), 1-리놀레오일-2-리놀레일옥시-3-디메틸아미노프로판 (DLin-2-DMAP), 1,2-디리놀레일옥시-3-트리메틸아미노프로판 염화물 염 (DLin-TMA.Cl), 1,2-디리놀레오일-3-트리메틸아미노프로판 염화물 염 (DLin-TAP.Cl), 1,2-디리놀레일옥시-3-(N-메틸피페라지노)프로판 (DLin-MPZ), 또는 3-(N,N-디리놀레일아미노)-1,2-프로판디올 (DLinAP), 3-(N,N-디올레일아미노)-1,2-프로판디오 (DOAP), 1,2-디리놀레일옥소-3-(2-N,N-디메틸아미노)에톡시프로판 (DLin-EG-DMA), 1,2-디리놀레닐옥시-N,N-디메틸아미노프로판 (DLinDMA), 2,2-디리놀레일-4-디메틸아미노메틸-[1,3]-디옥솔란 (DLin-K-DMA) 또는 이의 유사체, (3aR,5s,6aS)-N,N-디메틸-2,2-디((9Z,12Z)-옥타데카-9,12-디엔에테트라히드로-- 3aH-시클로펜타[d][1,3]디옥솔-5-아민 (ALN100), (6Z,9Z,28Z,31Z)-헵타트리아콘타-6,9,28,31-테트라엔-19-일4-(디메틸아미노)부타노에이트 (MC3), 1,1'-(2-(4-(2-((2-(비스(2-히드록시도데실)아미노)에틸)(2-히드록시도데실)아미노)에틸)피페라진-1-에에틸아잔디에디도데칸-2-올 (Tech G1), 또는 이들의 혼합물을 포함한다. 양이온성 지질은 예를 들면, 입자 내에 존재하는 총 지질의 약 20 mol % 내지 약 50 mol % 또는 약 40 mol %를 포함할 수 있다.
이온화가능/비양이온성 지질은 디스테아로일포스파티딜콜린 (DSPC), 디올레오일포스파티딜콜린 (DOPC), 디팔미토일포스파티딜콜린 (DPPC), 디올레오일포스파티딜글리세롤 (DOPG), 디팔미토일포스파티딜글리세롤 (DPPG), 디올레오일-포스파티딜에탄올아민 (DOPE), 팔미토일올레오일포스파티딜콜린 (POPC), 팔미토일올레오일포스파티딜에탄올아민 (POPE), 디올레오일-포스파티딜에탄올아민 4-(N-말레이미도메틸)-시클로헥산-1-카르복실산염 (DOPE-mal), 디팔미토일 포스파티딜 에탄올아민 (DPPE), 디미리스토일포스포에탄올아민 (DMPE), 디스테아로일-포스파티딜-에탄올아민 (DSPE), 16-O-모노메틸 PE, 16-O-디메틸 PE, 18-1-트랜스 PE, 1-스테아로일-2-올레오일-포스파티딜에탄올아민 (SOPE), 콜레스테롤, 또는 이들의 혼합물을 포함하지만 이들에 한정되지 않는 음이온성 지질 또는 중성 지질일 수 있다. 비양이온성 지질은 예를 들면, 만약 콜레스테롤이 포함되면, 입자 내에 존재하는 총 지질의 약 5 mol % 내지 약 90 mol %, 약 10 mol %, 또는 약 58 mol %일 수 있다.
입자의 응집을 저해하는 접합된 지질은 예를 들면, PEG-디아실글리세롤 (DAG), PEG-디알킬옥시프로필 (DAA), PEG-인지질, PEG-세라미드 (Cer), 또는 이들의 혼합물을 제한 없이 포함하는 폴리에틸렌글리콜 (PEG)-지질일 수 있다. PEG-DAA 접합체는 예를 들면, PEG-디라우릴옥시프로필 (Ci2), PEG-디미리스틸옥시프로필 (Ci4), PEG-디팔미틸옥시프로필 (Ci6), 또는 PEG-디스테아릴옥시프로필 (C]8)일 수 있다. 입자의 응집을 예방하는 접합된 지질은 예를 들면, 입자 내에 존재하는 총 지질의 0 mol % 내지 약 20 mol % 또는 약 2 mol %일 수 있다. 핵산-지질 입자는 예를 들면, 입자 내에 존재하는 총 지질의 약 10 mol % 내지 약 60 mol % 또는 약 50 mol %로 콜레스테롤을 더욱 포함할 수 있다.
리간드에 접합된 올리고뉴클레오티드
본원 발명의 올리고뉴클레오티드는 이러한 올리고뉴클레오티드의 활성, 세포 분포, 또는 세포 흡수를 증강하는 하나 이상의 리간드, 모이어티, 또는 접합체에 화학적으로 연결될 수 있다. 이런 모이어티는 지질 모이어티 예컨대 콜레스테롤 모이어티 (Letsinger et al., (1989) Proc. Natl. Acid. Sci. USA, 86: 6553-6556), 담즙산 (Manoharan et al., (1994) Biorg. Med. Chem. Let., 4:1053-1060), 티오에테르, 예를 들면, 베릴-S-트리틸티올 (Manoharan et al., (1992) Ann. N.Y. Acad. Sci., 660:306-309; Manoharan et al., (1993) Biorg. Med. Chem. Let., 3:2765-2770), 티오콜레스테롤 (Oberhauser et al., (1992) Nucl. Acids Res., 20:533-538), 지방족 사슬, 예를 들면, 도데칸디올 또는 운데실 잔기 (Saison-Behmoaras et al., (1991) EMBO J, 10:1111-1118; Kabanov et al., (1990) FEBS Lett., 259:327-330; Svinarchuk et al., (1993) Biochimie, 75:49-54), 인지질, 예를 들면, 디-헥사데실-rac-글리세롤 또는 트리에틸-암모늄 1,2-디-O-헥사데실-rac-글리세로-3-포스포네이트 (Manoharan et al., (1995) Tetrahedron Lett., 36:3651-3654; Shea et al., (1990) Nucl. Acids Res., 18:3777-3783), 폴리아민 또는 폴리에틸렌 글리콜 사슬 (Manoharan et al., (1995) Nucleosides & Nucleotides, 14:969-973), 또는 아다만탄 아세트산 (Manoharan et al., (1995) Tetrahedron Lett., 36:3651-3654), 팔미틸 모이어티 (Mishra et al., (1995) Biochim. Biophys. Acta, 1264:229-237), 또는 옥타데실아민 또는 헥실아미노-카르보닐옥시콜레스테롤 모이어티 (Crooke et al., (1996) J. Pharmacol. Exp. Ther., 277:923-937)를 포함하지만 이들에 한정되지 않는다.
리간드는 자신이 통합되는 올리고뉴클레오티드 작용제의 분포, 표적화 또는 수명을 변경할 수 있고 및/또는 예를 들면, 이런 리간드가 부재하는 종류와 비교하여, 선택된 표적, 예를 들면, 분자, 세포 또는 세포 유형, 구획, 예를 들면, 세포 또는 장기 구획, 조직, 장기, 또는 신체의 영역에 대한 증강된 친화성을 제공할 수 있다.
리간드는 자연 발생 물질, 예컨대 단백질 (예를 들면, 인간 혈청 알부민 (HSA), 저밀도 지질단백질 (LDL), 또는 글로불린); 탄수화물 (예를 들면, 덱스트란, 풀루란, 키틴, 키토산, 이눌린, 시클로덱스트린, N-아세틸글루코사민, N-아세틸갈락토사민, 또는 히알루론산); 또는 지질을 포함할 수 있다. 리간드는 또한, 재조합 또는 합성 분자, 예컨대 합성 중합체, 예를 들면, 합성 폴리아미노산일 수 있다. 폴리아미노산의 실례는 폴리리신 (PLL), 폴리 L-아스파르트산, 폴리 L-글루타민산, 스티렌-말레산 무수물 공중합체, 폴리(L-락티드-코-글리콜리드) 공중합체, 디비닐 에테르-말레산 무수물 공중합체, N-(2-히드록시프로필)메타크릴아미드 공중합체 (HMPA), 폴리에틸렌 글리콜 (PEG), 폴리비닐 알코올 (PVA), 폴리우레탄, 폴리(2-에틸아크릴산), N-이소프로필아크릴아미드 중합체, 또는 폴리포스파진과 같은 폴리아미노산을 포함한다. 폴리아민의 실례는 폴리에틸렌이민, 폴리리신 (PLL), 스페르민, 스페르미딘, 폴리아민, 슈도펩티드-폴리아민, 펩티드모방체 폴리아민, 덴드리머 폴리아민, 아르기닌, 아미딘, 프로타민, 양이온성 지질, 양이온성 포르피린, 폴리아민의 사차 염, 또는 알파 나선형 펩티드를 포함한다.
리간드는 또한, 표적화 기, 예를 들면, 세포 또는 조직 표적화 작용제, 예를 들면, 렉틴, 당단백질, 지질 또는 단백질, 예를 들면, 특정된 세포 유형 예컨대 신장 세포에 결합하는 항체를 포함할 수 있다. 표적화 기는 갑상선 자극 호르몬, 멜라노트로핀, 렉틴, 당단백질, 계면활성제 단백질 A, 점액소 탄수화물, 다가 락토오스, 다가 갈락토오스, N-아세틸-갈락토사민, N-아세틸-글루코사민 다가 만노오스, 다가 푸코오스, 글리코실화된 폴리아미노산, 다가 갈락토오스, 트랜스페린, 비스포스포네이트, 폴리글루타메이트, 폴리아스파르테이트, 지질, 콜레스테롤, 스테로이드, 담즙산, 엽산염, 비타민 B12, 비타민 A, 비오틴, 또는 RGD 펩티드 또는 RGD 펩티드 모방체일 수 있다.
리간드의 다른 실례는 염료, 중격제 (예를 들면 아크리딘), 교차연결제 (예를 들면 소랄렌, 미토마이신 C), 포르피린 (TPPC4, 텍사피린, 사피린), 다중환상 방향족 탄화수소 (예를 들면, 페나진, 디히드로페나진), 인공 엔도뉴클레아제 (예를 들면 EDTA), 친유성 분자, 예를 들면, 콜레스테롤, 담즙산, 아다만탄 아세트산, 1-피렌 부티르산, 디히드로테스토스테론, 1,3-비스-O(헥사데실)글리세롤, 제라닐옥시헥실 기, 헥사데실글리세롤, 보르네올, 멘톨, 1,3-프로판디올, 헵타데실 기, 팔미트산, 미리스트산, O3-(올레오일)리토콜산, O3-(올레오일)콜렌산, 디메톡시트리틸, 또는 페녹사진) 및 펩티드 접합체 (예를 들면, 안테나페디아 펩티드, Tat 펩티드), 알킬화제, 인산염, 아미노, 메르캅토, PEG (예를 들면, PEG-40K), MPEG, [MPEG]2, 폴리아미노, 알킬, 치환된 알킬, 방사성표지화된 마커, 효소, 합텐 (예를 들면 비오틴), 수송/흡수 촉진자 (예를 들면, 아스피린, 비타민 E, 엽산), 합성 리보핵산분해효소 (예를 들면, 이미다졸, 비스이미다졸, 히스타민, 이미다졸 클러스터, 아크리딘-이미다졸 접합체, 테트라아자마크로사이클의 Eu3+ 복합체), 디니트로페닐, HRP, 또는 AP를 포함한다.
리간드는 단백질, 예를 들면, 당단백질, 또는 펩티드, 예를 들면, 공동리간드에 대한 특이적 친화성을 갖는 분자, 또는 항체, 예를 들면, 특정된 세포 유형 예컨대 간 세포에 결합하는 항체일 수 있다. 리간드는 또한, 호르몬 및 호르몬 수용체를 포함할 수 있다. 이들은 또한, 비펩티드성 종류, 예컨대 지질, 렉틴, 탄수화물, 비타민, 보조인자, 다가 락토오스, 다가 갈락토오스, N-아세틸-갈락토사민, N-아세틸-글루코사민 다가 만노오스, 또는 다가 푸코오스를 포함할 수 있다.
리간드는 예를 들면, 세포의 세포골격을 파괴함으로써, 예를 들면, 세포의 미소관, 마이크로필라멘트 및/또는 중간 필라멘트를 파괴함으로써, 올리고뉴클레오티드 작용제의 세포 내로의 흡수를 증가시킬 수 있는 물질, 예를 들면, 약물일 수 있다. 약물은 예를 들면 탁손, 빈크리스틴, 빈블라스틴, 시토칼라신, 노코다졸, 자스플라키놀리드, 라트룬쿨린 A, 팔로이딘, 스윈홀리드 A, 인다노신, 또는 미오서빈일 수 있다.
본원에서 설명된 바와 같은 올리고뉴클레오티드에 부착된 리간드는 약물동력학적 조절인자 (PK 조절인자)로서 작용할 수 있다. PK 조절인자는 친유성기, 담즙산, 스테로이드, 인지질 유사체, 펩티드, 단백질 결합 작용제, PEG, 비타민 등을 포함한다. 예시적인 PK 조절인자는 콜레스테롤, 지방산, 담즙산, 리토콜산, 디알킬글리세리드, 디아실글리세리드, 인지질, 스핑고지질, 나프록센, 이부프로펜, 비타민 E, 비오틴 등을 포함하지만 이들에 한정되지 않는다. 다수의 포스포로티오에이트 연쇄를 포함하는 올리고뉴클레오티드는 또한, 혈청 단백질에 결합하는 것으로 알려져 있고, 따라서 백본 내에 복수의 포스포로티오에이트 연쇄를 포함하는 짧은 올리고뉴클레오티드, 예를 들면, 약 5개 염기, 10개 염기, 15개 염기, 또는 20개 염기의 올리고뉴클레오티드 또한, 리간드로서 (예를 들면 PK 조정 리간드로서) 본원 발명에 적합하다. 이에 더하여, 혈청 성분 (예를 들면 혈청 단백질)에 결합하는 앱타머 또한, 본원에서 설명된 방법과 조성물에서 PK 조정 리간드로서 이용에 적합하다.
본원 발명의 리간드-접합된 올리고뉴클레오티드는 펜던트 반응성 기능기, 예컨대 올리고뉴클레오티드 위에 연결 분자의 부착으로부터 유래된 것을 보유하는 올리고뉴클레오티드의 이용에 의해 합성될 수 있다 (아래에 설명됨). 이러한 반응성 올리고뉴클레오티드는 상업적으로 가용한 리간드, 임의의 다양한 보호기를 보유하는 합성되는 리간드, 또는 연결 모이어티가 부착된 리간드와 직접적으로 반응될 수 있다.
본원 발명의 접합체에서 이용되는 올리고뉴클레오티드는 고체상 합성의 널리 알려진 기술을 통해 편의하게 및 일과적으로 만들어질 수 있다. 이런 합성을 위한 설비는 예를 들면, Applied Biosystems (Foster City, Calif.)를 비롯한, 여러 판매자에 의해 판매된다. 당해 분야에서 알려진 이런 합성을 위한 임의의 다른 수단이 부가적으로 또는 대안적으로 이용될 수 있다. 유사한 기술을 이용하여 다른 올리고뉴클레오티드, 예컨대 포스포로티오에이트 및 알킬화된 유도체를 제조하는 것 또한 알려져 있다.
본원 발명의 리간드-접합된 올리고뉴클레오티드, 예컨대 본원 발명의 리간드-분자 보유 서열 특이적 연결된 뉴클레오시드에서, 올리고뉴클레오티드 및 올리고뉴클레오시드는 표준 뉴클레오티드 또는 뉴클레오시드 전구체, 또는 연결 모이어티를 이미 보유하는 뉴클레오티드 또는 뉴클레오시드 접합체 전구체, 리간드 분자를 이미 보유하는 리간드-뉴클레오티드 또는 뉴클레오시드-접합체 전구체, 또는 비뉴클레오시드 리간드-보유 빌딩 블록을 활용하는 적합한 DNA 합성장치에서 조립될 수 있다.
연결 모이어티를 이미 보유하는 뉴클레오티드-접합체 전구체를 이용할 때, 서열 특이적 연결된 뉴클레오시드의 합성이 전형적으로 완료되고, 그리고 리간드 분자가 이후, 연결 모이어티와 반응되어 리간드-접합된 올리고뉴클레오티드가 형성된다. 본원 발명의 올리고뉴클레오티드 또는 연결된 뉴클레오시드는, 상업적으로 가용하고 올리고뉴클레오티드 합성에서 일과적으로 이용되는 표준 포스포라미디트 및 비표준 포스포라미디트에 더하여, 리간드-뉴클레오시드 접합체로부터 유래된 포스포라미디트를 이용한 자동화 합성장치에 의해 합성될 수 있다.
i. 지질 접합체
본원 발명에 따라서, 리간드 또는 접합체는 지질 또는 지질 기반 분자일 수 있다. 이런 지질 또는 지질 기반 분자는 혈청 단백질, 예를 들면, 인간 혈청 알부민 (HSA)에 특이적으로 결합한다. HSA 결합 리간드는 신체의 표적 조직, 예를 들면, 비-신장 표적 조직에 접합체의 분포를 가능하게 한다. 예를 들면, 표적 조직은 간 (간의 실질성 세포 포함)일 수 있다. HSA에 결합할 수 있는 다른 분자 역시 리간드로서 이용될 수 있다. 예를 들면, 나프록센 또는 아스피린이 이용될 수 있다. 지질 또는 지질 기반 리간드는 (a) 접합체의 분해에 대한 내성을 증가시킬 수 있고, (b) 표적 세포 또는 세포막 내로의 표적화 또는 수송을 증가시킬 수 있고, 및/또는 (c) 혈청 단백질, 예를 들면, HSA에 결합을 조정하는 데 이용될 수 있다.
지질 기반 리간드는 표적 조직에 대한 접합체의 결합을 저해하는, 예를 들면, 제어하는 데 이용될 수 있다. 예를 들면, HSA에 더 강하게 결합하는 지질 또는 지질 기반 리간드는 신장으로 표적화될 가능성이 더 낮을 것이고, 그리고 이런 이유로 신체로부터 소실될 가능성이 더 낮을 것이다. HSA에 덜 강하게 결합하는 지질 또는 지질 기반 리간드는 접합체를 신장으로 표적화하는 데 이용될 수 있다.
다른 양상에서, 리간드는 표적 세포, 예를 들면, 증식 세포에 의해 흡수되는 모이어티, 예를 들면, 비타민일 수 있다. 예시적인 비타민은 비타민 A, E 및 K를 포함한다.
ii. 세포 투과 작용제
리간드는 또한, 세포 투과 작용제, 예를 들면, 나선형 세포 투과 작용제일 수 있다. 특정 실례에서, 작용제는 양친매성이다. 예시적인 작용제는 펩티드 예컨대 tat 또는 안테나페디아이다. 만약 작용제가 펩티드이면, 이것은 펩티딜모방체, 인베르토머, 비펩티드 또는 슈도펩티드 연쇄, 그리고 D-아미노산의 이용을 비롯하여, 변형될 수 있다. 나선 작용제는 친유성 및 소유성 상을 갖는 알파-나선 작용제일 수 있다.
리간드는 펩티드 또는 펩티드모방체일 수 있다. 펩티드모방체 (본원에서 올리고펩티드모방체로서 또한 지칭됨)는 천연 펩티드와 유사한 규정된 3차원 구조로 접힐 수 있는 분자이다. 올리고뉴클레오티드 작용제에 펩티드 및 펩티드모방체의 부착은 예컨대 세포 인식 및 흡수를 증강함으로써, 올리고뉴클레오티드의 약물동력학적 분포에 영향을 줄 수 있다. 펩티드 또는 펩티드모방체 모이어티는 약 5-50개 아미노산 길이, 예를 들면, 약 5, 10, 15, 20, 25, 30, 35, 40, 45, 또는 50개 아미노산 길이일 수 있다.
펩티드 또는 펩티드모방체는 예를 들면, 세포 투과 펩티드, 양이온성 펩티드, 양친매성 펩티드, 또는 소수성 펩티드 (예를 들면, Tyr, Trp 또는 Phe로 주로 구성됨)일 수 있다. 펩티드 모이어티는 덴드리머 펩티드, 제약된 펩티드 또는 교차연결된 펩티드일 수 있다. 다른 대안에서, 펩티드 모이어티는 소수성 막 전위 서열 (MTS)을 포함할 수 있다. 예시적인 소수성 MTS 내포 펩티드는 아미노산 서열 AAVALLPAVLLALLAP를 갖는 RFGF이다. RFGF 유사체 (예를 들면, 소수성 MTS를 내포하는 아미노산 서열 AALLPVLLAAP 또한 표적화 모이어티일 수 있다). 펩티드 모이어티는 펩티드, 올리고뉴클레오티드 및 단백질을 비롯한 큰 극성 분자를 세포막을 교차하여 운반할 수 있는 "전달" 펩티드일 수 있다. 예를 들면, HIV Tat 단백질 (GRKKRRQRRRPPQ) 및 초파리 (Drosophila) 안테나페디아 (Antennapedia) 단백질 (RQIKIWFQNRRMKWKK)로부터 유래된 서열이 전달 펩티드로서 기능할 수 있는 것으로 밝혀졌다. 펩티드 또는 펩티드모방체는 DNA의 무작위 서열에 의해 인코딩될 수 있다, 예컨대 파지 전시 라이브러리, 또는 1-비드-1-화합물 (OBOC) 조합 라이브러리로부터 확인된 펩티드일 수 있다 (Lam et al., Nature, 354:82-84, 1991). 세포 표적화 목적으로, 통합된 단량체 단위를 통해 올리고뉴클레오티드 작용제에 묶인 펩티드 또는 펩티드모방체의 실례는 아르기닌-글리신-아스파르트산 (RGD)-펩티드, 또는 RGD 모방체이다. 펩티드 모이어티는 길이에서 약 5개 아미노산 내지 약 40개 아미노산의 범위 안에 있을 수 있다. 펩티드 모이어티는 예컨대 안정성을 증가시키거나 또는 입체형태적 특성을 주도하기 위한 구조적 변형을 가질 수 있다. 아래에 설명된 임의의 구조적 변형이 활용될 수 있다.
RGD 펩티드는 조성물을 세포 표적으로 지향시키기 위해 본원 발명의 조성물에서 이용될 수 있다. RGD 펩티드는 선형 또는 환상일 수 있고, 그리고 특정한 조직(들)으로의 표적화를 가능하게 하기 위해 변형될 수 있다, 예를 들면, 글리코실화 또는 메틸화될 수 있다. RGD 내포 펩티드 및 펩티드모방체는 D-아미노산뿐만 아니라 합성 RGD 모방체를 포함할 수 있다. RGD에 더하여, 인테그린 리간드를 표적으로 하는 다른 모이어티가 이용될 수 있다. 이러한 리간드의 일부 접합체는 PECAM-1 또는 VEGF를 표적으로 한다.
세포 투과 펩티드는 세포, 예를 들면, 미생물 세포, 예컨대 세균 또는 진균 세포, 또는 포유류 세포, 예컨대 인간 세포를 침투할 수 있다. 미생물 세포 침투 펩티드는 예를 들면, α-나선 선형 펩티드 (예를 들면, LL-37 또는 세로핀 P1), 이황화 결합 내포 펩티드 (예를 들면, α-디펜신, β-디펜신 또는 박테네신), 또는 단지 1개 또는 2개의 우세한 아미노산을 내포하는 펩티드 (예를 들면, PR-39 또는 인돌리시딘)일 수 있다. 세포 투과 펩티드는 또한, 핵 국지화 신호 (NLS)를 포함할 수 있다. 예를 들면, 세포 투과 펩티드는 HIV-1 gp41의 융합 펩티드 도메인 및 SV40 큰 T 항원의 NLS로부터 유래되는 양분 양친매성 펩티드, 예컨대 MPG일 수 있다 (Simeoni et al., Nucl. Acids Res. 31:2717-2724, 2003).
iii. 탄수화물 접합체
본원 발명의 조성물과 방법에 따라서, 올리고뉴클레오티드는 탄수화물을 더욱 포함할 수 있다. 탄수화물 접합된 올리고뉴클레오티드는 본원에서 설명된 바와 같이, 핵산뿐만 아니라 생체내 치료적 용도에 적합한 조성물의 생체내 전달에 유리하다. 본원에서 이용된 바와 같이, "탄수화물"은 적어도 6개의 탄소 원자 (이들은 선형, 분지형 또는 환상일 수 있다)를 갖고 산소, 질소 또는 황 원자가 각 탄소 원자에 결합된 하나 이상의 단당류 단위로 구성되는 탄수화물 그 자체인 화합물; 또는 적어도 6개의 탄소 원자 (이들은 선형, 분지형 또는 환상일 수 있다)를 각각 갖고 산소, 질소 또는 황 원자가 각 탄소 원자에 결합된 하나 이상의 단당류 단위로 구성되는 탄수화물 모이어티를 자신의 일부로서 갖는 화합물을 지칭한다. 대표적인 탄수화물은 당 (약 4, 5, 6, 7, 8 또는 9개의 단당류 단위를 내포하는 모노-, 디-, 트리- 및 올리고당류), 그리고 다당류 예컨대 전분, 글리코겐, 셀룰로오스 및 다당류 검을 포함한다. 특정한 단당류는 C5 이상 (예를 들면, C5, C6, C7 또는 C8) 당을 포함하고; 이당류 및 삼당류는 2개 또는 3개의 단당류 단위를 갖는 당 (예를 들면, C5, C6, C7 또는 C8)을 포함한다.
특정 실례에서, 본원 발명의 조성물과 방법에 이용하기 위한 탄수화물 접합체는 단당류이다. 탄수화물 접합체는 전술된 바와 같은 하나 이상의 추가 리간드, 예컨대 그러나 제한 없이, PK 조절인자 및/또는 세포 투과 펩티드를 더욱 포함할 수 있다. 본원 발명에 이용하기 적합한 추가의 탄수화물 접합체 (및 링커)는 PCT 공개 번호 WO 2014/179620 및 WO 2014/179627에서 설명된 것들을 포함하는데, 이들 각각의 전체 내용은 본원에서 참조로서 편입된다.
iv. 링커
본원에서 설명된 접합체 또는 리간드는 개열 가능하거나 또는 비개열 가능할 수 있는 다양한 링커로 올리고뉴클레오티드에 부착될 수 있다.
링커는 전형적으로, 직접적인 결합 또는 원자 예컨대 산소 또는 황, 단위 예컨대 NR8, C(O), C(O)NH, SO, SO2, SO2NH 또는 원자의 사슬, 예컨대 그러나 제한 없이, 치환된 또는 치환되지 않은 알킬, 치환된 또는 치환되지 않은 알케닐, 치환된 또는 치환되지 않은 알키닐, 아릴알킬, 아릴알케닐, 아릴알키닐, 헤테로아릴알킬, 헤테로아릴알케닐, 헤테로아릴알키닐, 헤테로시클릴알킬, 헤테로시클릴알케닐, 헤테로시클릴알키닐, 아릴, 헤테로아릴, 헤테로시클릴, 시클로알킬, 시클로알케닐, 알킬아릴알킬, 알킬아릴알케닐, 알킬아릴알키닐, 알케닐아릴알킬, 알케닐아릴알케닐, 알케닐아릴알키닐, 알키닐아릴알킬, 알키닐아릴알케닐, 알키닐아릴알키닐, 알킬헤테로아릴알킬, 알킬헤테로아릴알케닐, 알킬헤테로아릴알키닐, 알케닐헤테로아릴알킬, 알케닐헤테로아릴알케닐, 알케닐헤테로아릴알키닐, 알키닐헤테로아릴알킬, 알키닐헤테로아릴알케닐, 알키닐헤테로아릴알키닐, 알킬헤테로시클릴알킬, 알킬헤테로시클릴알케닐, 알킬헤테로시클릴알키닐, 알케닐헤테로시클릴알킬, 알케닐헤테로시클릴알케닐, 알케닐헤테로시클릴알키닐, 알키닐헤테로시클릴알킬, 알키닐헤테로시클릴알케닐, 알키닐헤테로시클릴알키닐, 알킬아릴, 알케닐아릴, 알키닐아릴, 알킬헤테로아릴, 알케닐헤테로아릴, 알키닐헤테로아릴을 포함하고, 여기서 하나 이상의 메틸렌이 O, S, S(O), SO2, N(R8), C(O), 치환된 또는 치환되지 않은 아릴, 치환된 또는 치환되지 않은 헤테로아릴, 치환된 또는 치환되지 않은 헤테로환상에 의해 중단되거나 또는 종결될 수 있고; 여기서 R8은 수소, 아실, 지방족 또는 치환된 지방족이다. 특정 실례에서, 링커는 약 1-24개의 원자, 2-24, 3-24, 4-24, 5-24, 6-24, 6-18, 7-18, 8-18개의 원자, 7-17, 8-17, 6-16, 7-17, 또는 8-16개의 원자 사이일 수 있다.
개열가능 연결기는 세포 외부에서는 충분히 안정되지만 표적 세포 내로 진입 시에 개열되어, 링커가 결속시키고 있는 두 부분을 방출하는 것이다. 바람직한 구체예에서, 개열가능 연결기는 표적 세포에서, 또는 개체의 혈액에서보다 첫 번째 참조 조건 (이것은 예를 들면, 세포내 조건을 모의하거나 또는 나타내도록 선택될 수 있다)하에, 또는 두 번째 참조 조건 (이것은 예를 들면, 혈액 또는 혈청에서 발견되는 조건을 모의하거나 또는 나타내도록 선택될 수 있다)하에, 적어도 약 10 배, 20 배, 30 배, 40 배, 50 배, 60 배, 70 배, 80 배, 90 배, 또는 그 초과, 또는 적어도 약 100 배 빨리 개열된다.
개열가능 연결기는 개열 작용제, 예를 들면, pH, 산화환원 전위, 또는 분해성 분자의 존재에 대해 감수성이다. 일반적으로, 개열 작용제는 혈청 또는 혈액에서보다 세포 내부에서 더 우세하거나, 또는 더 높은 수준 또는 활성으로 발견된다. 이런 분해성 작용제의 실례는 예를 들면, 환원에 의해 산화환원 개열가능 연결기를 분해할 수 있는, 세포 내에 존재하는 산화 또는 환원 효소 또는 환원성 작용제 예컨대 메르캅탄을 비롯한, 특정 기질에 대해 선택적이거나 또는 기질 특이성이 없는 산화환원 작용제; 에스테라아제; 엔도솜 또는 산성 환경을 창출할 수 있는 작용제, 예를 들면, 5 이하의 pH를 야기하는 것들; 일반 산으로서 작용함으로써 산 개열가능 연결기를 가수분해하거나 또는 분해할 수 있는 효소, 펩티드분해효소 (기질 특이적일 수 있다), 그리고 포스파타아제를 포함한다.
개열가능 연결기, 예컨대 이황화 결합은 pH에 대해 감수성일 수 있다. 인간 혈청의 pH는 7.4이고, 반면 평균 세포내 pH는 약간 더 낮은, 약 7.1-7.3의 범위 안에 있다. 엔도솜은 5.5-6.0 범위의 더 높은 산성 pH를 갖고, 그리고 리소좀은 대략 5.0의 훨씬 높은 산성 pH를 갖는다. 일부 링커는, 바람직한 pH에서 개열되고, 그것에 의하여 양이온성 지질을 리간드로부터 세포 내부에, 또는 세포의 원하는 구획 내로 방출하는 개열가능 연결기를 가질 것이다.
링커는 특정 효소에 의해 개열 가능한 개열가능 연결기를 포함할 수 있다. 링커 내로 통합된 개열가능 연결기의 유형은 표적화되는 세포에 의존할 수 있다. 예를 들면, 간 표적화 리간드가 에스테르 기를 포함하는 링커를 통해 양이온성 지질에 연결될 수 있다. 간 세포는 에스테라아제가 풍부하고, 그리고 이런 이유로 링커는 에스테라아제가 풍부하지 않는 세포 유형에서보다 간 세포에서 더 효율적으로 개열될 것이다. 에스테라아제가 풍부한 다른 세포 유형은 폐, 신장 피질 및 고환의 세포를 포함한다.
펩티드 결합을 내포하는 링커는 펩티드분해효소가 풍부한 세포 유형, 예컨대 간 세포 및 윤활막세포를 표적으로 할 때 이용될 수 있다.
일반적으로, 후보 개열가능 연결기의 적합성은 후보 연결기를 개열하는 분해성 작용제 (또는 조건)의 능력을 검사함으로써 평가될 수 있다. 혈액 내에서 또는 다른 비표적 조직과 접촉할 때 개열에 저항하는 능력에 대해 후보 개열가능 연결기를 또한 검사하는 것이 또한 바람직할 것이다. 따라서, 첫 번째와 두 번째 조건 사이에 개열에 대한 상대적 감수성이 결정될 수 있는데, 여기서 첫 번째 조건은 표적 세포에서 개열을 표시하도록 선택되고, 그리고 두 번째 조건은 다른 조직 또는 생물학적 유체, 예를 들면, 혈액 또는 혈청에서 개열을 표시하도록 선택된다. 평가는 무세포 시스템에서, 세포에서, 세포 배양액에서, 장기 또는 조직 배양액에서, 또는 전체 동물에서 실행될 수 있다. 이것은 무세포 또는 배양 조건에서 초기 평가를 하고 전체 동물에서 추가 평가에 의해 확증하는 데 유용할 수 있다. 일부 경우에, 유용한 후보 화합물은 혈액 또는 혈청 (또는 세포외 조건을 모의하도록 선택된 시험관내 조건하에)에서와 비교하여 세포 (또는 세포내 조건을 모의하도록 선택된 시험관내 조건하에)에서 적어도 약 2, 4, 10, 20, 30, 40, 50, 60, 70, 80, 90, 또는 약 100 배 빨리 개열된다.
a. 산화환원 개열가능 연결기
개열가능 연결기는 환원 또는 산화 시에 개열되는 산화환원 개열가능 연결기일 수 있다. 환원성으로 개열가능 연결기의 실례는 이황화물 연결기 (--S--S--)이다. 후보 개열가능 연결기가 적합한 "환원성으로 개열가능 연결기"인지를 또는 예를 들면, 특정 올리고뉴클레오티드 모이어티 및 특정 표적화 작용제용으로 적합한지를 결정하기 위해, 본원에서 설명된 방법이 고려될 수 있다. 예를 들면, 세포, 예를 들면, 표적 세포에서 관찰될 개열 속도를 모방하는, 당해 분야에서 공지된 시약을 이용하여, 디티오에리트리톨 (DTE) 또는 다른 환원제와 함께 인큐베이션에 의해, 후보가 평가될 수 있다. 후보는 또한, 혈액 또는 혈청 조건을 모의하도록 선택되는 조건하에 평가될 수 있다. 후보 화합물은 혈액 내에서 약 10% 이하로 개열될 수 있다. 다른 실례에서, 유용한 후보 화합물은 혈액 (또는 세포외 조건을 모의하도록 선택된 시험관내 조건하에)에서와 비교하여 세포 (또는 세포내 조건을 모의하도록 선택된 시험관내 조건하에)에서 적어도 약 2, 4, 10, 20, 30, 40, 50, 60, 70, 80, 90, 또는 약 100 배 빨리 분해된다. 후보 화합물의 개열 속도는 세포내 배지를 모의하도록 선택된 조건하에 표준 효소 동역학 검정을 이용하여 결정되고 세포외 배지를 모의하도록 선택된 조건과 비교될 수 있다.
b. 인산염 기반 개열가능 연결기
개열가능 링커는 또한, 인산염 기반 개열가능 연결기를 포함할 수 있다. 인산염 기반 개열가능 연결기는 인산염 기를 분해하거나 또는 가수분해하는 작용제에 의해 개열된다. 세포에서 인산염 기를 개열하는 작용제의 실례는 세포에서 포스파타아제와 같은 효소이다. 인산염 기반 연결기의 실례는 -O-P(O)(ORk)-O-,
-O-P(S)(ORk)-O-, -O-P(S)(SRk)-O-, -S-P(O)(ORk)-O-, -O-P(O)(ORk)-S-, -S-P(O)(ORk)-S-,
-O-P(S)(ORk)-S-, -S-P(S)(ORk)-O-, -O-P(O)(Rk)-O-, -O-P(S)(Rk)-O-, -S-P(O)(Rk)-O-, -S-P(S)(Rk)-O-,
-S-P(O)(Rk)-S-, -O-P(S)(Rk)-S-이다. 이들 후보는 전술된 것들과 유사한 방법을 이용하여 평가될 수 있다.
c. 산 개열가능 연결기
개열가능 링커는 또한, 산 개열가능 연결기를 포함할 수 있다. 산 개열가능 연결기는 산성 조건하에 개열되는 연결기이다. 바람직한 구체예에서 산 개열가능 연결기는 약 6.5 또는 그 미만 (예를 들면, 약 6.0, 5.75, 5.5, 5.25, 5.0, 또는 그 미만)의 pH를 갖는 산성 환경에서, 또는 일반 산으로서 작용할 수 있는 작용제 예컨대 효소에 의해 개열된다. 세포에서, 특정한 낮은 pH 소기관, 예컨대 엔도솜 및 리소좀이 산 개열가능 연결기에 대한 개열 환경을 제공할 수 있다. 산 개열가능 연결기의 실례는 히드라존, 에스테르, 그리고 아미노산의 에스테르를 포함하지만 이들에 한정되지 않는다. 산 개열가능 기는 일반식 -C=NN--, C(O)O, 또는 --OC(O)를 가질 수 있다. 바람직한 구체예는 에스테르의 산소 (알콕시 기)에 부착된 탄소가 아릴 기, 치환된 알킬 기, 또는 삼차 알킬 기 예컨대 디메틸 펜틸 또는 t-부틸일 때이다. 이들 후보는 전술된 것들과 유사한 방법을 이용하여 평가될 수 있다.
d. 에스테르 기반 연결기
개열가능 링커는 에스테르 기반 개열가능 연결기를 포함할 수 있다. 에스테르 기반 개열가능 연결기는 세포에서 에스테라아제 및 아미다아제와 같은 효소에 의해 개열된다. 에스테르 기반 개열가능 연결기의 실례는 알킬렌, 알케닐렌 및 알키닐렌 기의 에스테르를 포함하지만 이들에 한정되지 않는다. 에스테르 개열가능 연결기는 일반식 --C(O)O--, 또는 --OC(O)--를 갖는다. 이들 후보는 전술된 것들과 유사한 방법을 이용하여 평가될 수 있다.
e. 펩티드 기반 개열 기
개열가능 링커는 펩티드 기반 개열가능 연결기를 더욱 포함할 수 있다. 펩티드 기반 개열가능 연결기는 세포에서 펩티드분해효소 및 프로테아제와 같은 효소에 의해 개열된다. 펩티드 기반 개열가능 연결기는 올리고펩티드 (예를 들면, 디펩티드, 트리펩티드 등) 및 폴리펩티드를 산출하는, 아미노산 사이에 형성된 펩티드 결합이다. 펩티드 기반 개열가능 기는 아미드 기 (--C(O)NH--)를 포함하지 않는다. 아미드 기는 임의의 알킬렌, 알케닐렌, 또는 알키넬렌 사이에 형성될 수 있다. 펩티드 결합은 펩티드와 단백질을 산출하는, 아미노산 사이에 형성된 특수한 유형의 아미드 결합이다. 펩티드 기반 개열 기는 일반적으로, 펩티드와 단백질을 산출하는, 아미노산 사이에 형성된 펩티드 결합 (다시 말하면, 아미드 결합)에 한정되고 전체 아미드 기능기를 포함하지 않는다. 펩티드 기반 개열가능 연결기는 일반식 --NHCHRAC(O)NHCHRBC(O)--를 갖는데, 여기서 RA 및 RB는 2개의 인접한 아미노산의 R 기이다. 이들 후보는 전술된 것들과 유사한 방법을 이용하여 평가될 수 있다.
본원 발명의 올리고뉴클레오티드는 링커를 통해 탄수화물에 접합될 수 있다. 링커는 이가 및 삼가 분지된 링커 기를 포함한다. 본원 발명의 조성물과 방법의 링커와의 예시적인 올리고뉴클레오티드 탄수화물 접합체는 PCT 공개 번호 WO 2018/195165의 화학식 24-35에서 설명된 것들을 포함하지만 이들에 한정되지 않는다.
올리고뉴클레오티드 접합체의 제조를 교시하는 대표적인 U.S. 특허는 U.S. 특허 번호 4,828,979; 4,948,882; 5,218,105; 5,525,465; 5,541,313; 5,545,730; 5,552,538; 5,578,717, 5,580,731; 5,591,584; 5,109,124; 5,118,802; 5,138,045; 5,414,077; 5,486,603; 5,512,439; 5,578,718; 5,608,046; 4,587,044; 4,605,735; 4,667,025; 4,762,779; 4,789,737; 4,824,941; 4,835,263; 4,876,335; 4,904,582; 4,958,013; 5,082,830; 5,112,963; 5,214,136; 5,082,830; 5,112,963; 5,214,136; 5,245,022; 5,254,469; 5,258,506; 5,262,536; 5,272,250; 5,292,873; 5,317,098; 5,371,241, 5,391,723; 5,416,203, 5,451,463; 5,510,475; 5,512,667; 5,514,785; 5,565,552; 5,567,810; 5,574,142; 5,585,481; 5,587,371; 5,595,726; 5,597,696; 5,599,923; 5,599,928 및 5,688,941; 6,294,664; 6,320,017; 6,576,752; 6,783,931; 6,900,297; 7,037,646; 8,106,022를 포함하지만 이들에 한정되지 않고, 이들의 전체 내용은 각각 본원에서 참조로서 편입된다.
일정한 사례에서, 올리고뉴클레오티드의 뉴클레오티드는 비-리간드 기에 의해 변형될 수 있다. 올리고뉴클레오티드의 활성, 세포 분포, 또는 세포 흡수를 증강하기 위해 다수의 비-리간드 분자가 올리고뉴클레오티드에 접합되었고, 그리고 이런 접합을 수행하기 위한 절차는 과학 문헌에서 가용하다. 이런 비-리간드 모이어티는 지질 모이어티, 예컨대 콜레스테롤 (Kubo, T. et al., Biochem. Biophys. Res. Comm, 2007, 365(1):54-61; Letsinger et al., Proc. Natl. Acad. Sci. USA, 1989, 86:6553), 담즙산 (Manoharan et al., Bioorg. Med. Chem. Lett., 1994, 4:1053), 티오에테르, 예를 들면, 헥실-S-트리틸티올 (Manoharan et al., Ann. N.Y. Acad. Sci., 1992, 660:306; Manoharan et al., Bioorg. Med. Chem. Let., 1993, 3:2765), 티오콜레스테롤 (Oberhauser et al., Nucl. Acids Res., 1992, 20:533), 지방족 사슬, 예를 들면, 도데칸디올 또는 운데실 잔기 (Saison-Behmoaras et al., EMBO J., 1991, 10:111; Kabanov et al., FEBS Lett., 1990, 259:327; Svinarchuk et al., Biochimie, 1993, 75:49), 인지질, 예를 들면, 디-헥사데실-rac-글리세롤 또는 트리에틸암모늄 1,2-디-O-헥사데실-rac-글리세로-3-H-포스포네이트 (Manoharan et al., Tetrahedron Lett., 1995, 36:3651; Shea et al., Nucl. Acids Res., 1990, 18:3777), 폴리아민 또는 폴리에틸렌 글리콜 사슬 (Manoharan et al., Nucleosides & Nucleotides, 1995, 14:969), 또는 아다만탄 아세트산 (Manoharan et al., Tetrahedron Lett., 1995, 36:3651), 팔미틸 모이어티 (Mishra et al., Biochim. Biophys. Acta, 1995, 1264:229), 또는 옥타데실아민 또는 헥실아미노-카르보닐-옥시콜레스테롤 모이어티 (Crooke et al., J. Pharmacol. Exp. Ther., 1996, 277:923)를 포함하였다. 이런 올리고뉴클레오티드 접합체의 제조를 교시하는 대표적인 미국 특허는 상기 열거되었다. 전형적인 접합 프로토콜은 서열의 하나 이상의 위치에서 아미노 링커를 보유하는 올리고뉴클레오티드의 합성을 수반한다. 아미노 기는 이후, 적합한 연계 또는 활성화 시약을 이용하여 접합되는 분자와 반응된다. 접합 반응은 올리고뉴클레오티드가 고체 지지체에 여전히 결합된 상태로, 또는 올리고뉴클레오티드의 개열 이후에, 용액상에서 수행될 수 있다. HPLC에 의한 올리고뉴클레오티드 접합체의 정제는 전형적으로, 순수한 접합체를 제공한다.
핵산 벡터
본원에서 개시된 핵산 작용제의 효과적인 세포내 농도는 상기 작용제를 인코딩하는 폴리뉴클레오티드의 안정된 발현 (예를 들면, 포유류 세포의 핵 또는 미토콘드리아 유전체 내로 통합에 의한)을 통해 달성될 수 있다. 핵산은 Grik2 mRNA를 표적으로 하는 저해성 RNA (예를 들면, 본원에서 개시된 ASO 작용제)이다. 이런 외인성 핵산을 포유류 세포 내로 도입하기 위해, 상기 작용제에 대한 폴리뉴클레오티드 서열이 벡터 내로 통합될 수 있다. 벡터는 형질전환, 형질감염, 직접적인 흡수, 발사체 폭격, 그리고 리포솜 내에 벡터의 캡슐화를 비롯한, 다양한 방법에 의해 세포 내로 도입될 수 있다. 세포를 형질감염시키거나 또는 형질전환하는 적합한 방법의 실례는 인산칼슘 침전, 전기천공, 미세주입, 감염, 리포펙션, 그리고 직접적인 흡수이다. 이런 방법은 예를 들면, Green et al., Molecular Cloning: A Laboratory Manual, Fourth Edition (Cold Spring Harbor University Press, New York (2014)); 및 Ausubel et al., Current Protocols in Molecular Biology (John Wiley & Sons, New York (2015))에서 더 상세하게 설명되는데, 이들 각각의 개시는 본원에서 참조로서 편입된다.
본원에서 개시된 작용제는 또한, 이런 작용제를 인코딩하는 폴리뉴클레오티드를 내포하는 벡터를 세포막 인지질로 표적화함으로써 포유류 세포 내로 도입될 수 있다. 예를 들면, 벡터는 벡터 분자를, 모든 세포막 인지질에 대한 친화성을 갖는 바이러스 단백질인 VSV-G 단백질에 연결함으로써 세포막의 세포외 표면 상에서 인지질에 표적화될 수 있다. 이런 작제물은 당해 분야의 전통적이고 일과적인 방법을 이용하여 생산될 수 있다.
높은 비율의 전사와 번역을 달성하는 것에 더하여, 포유류 세포에서 외인성 폴리뉴클레오티드의 안정된 발현이, 유전자를 내포하는 폴리뉴클레오티드를 포유류 세포의 핵 유전체 내로 통합함으로써 달성될 수 있다. 외인성 단백질을 인코딩하는 폴리뉴클레오티드의 포유류 세포의 핵 DNA 내로의 전달과 통합을 위한 다양한 벡터가 개발되었다. 발현 벡터의 실례는 예를 들면, WO 1994/011026에서 개시되고 본원에서 참조로서 편입된다. 본원에서 설명된 조성물과 방법에 이용하기 위한 발현 벡터는 Grik2 표적화 ASO 작용제를 인코딩하는 폴리뉴클레오티드 서열뿐만 아니라, 예를 들면, 이들 작용제의 발현 및/또는 이들 폴리뉴클레오티드 서열의 포유류 세포의 유전체 내로의 통합에 이용되는 추가의 서열 요소를 내포한다. 이용될 수 있는 일정한 벡터는 유전자 전사를 주도하는 조절 서열, 예컨대 프로모터와 인핸서 영역을 내포하는 플라스미드를 포함한다. 다른 유용한 벡터는 이들 유전자의 번역 속도를 증강하거나 또는 유전자 전사로부터 발생하는 mRNA의 안정성 또는 핵 이출을 향상시키는 폴리뉴클레오티드 서열을 내포한다. 이들 서열 요소는 예를 들면, 발현 벡터 상에서 운반되는 유전자의 효율적인 전사를 주도하기 위해 5' 및 3' UTR 영역, IRES, 그리고 폴리아데닐화 신호 부위를 포함한다. 본원에서 설명된 조성물과 방법용으로 적합한 발현 벡터는 또한, 이런 벡터를 내포하는 세포의 선택을 위한 마커를 인코딩하는 폴리뉴클레오티드를 내포할 수 있다. 적합한 마커의 실례는 항생제, 예컨대 암피실린, 클로람페니콜, 카나마이신, 누르세오트리신에 대한 내성을 인코딩하는 유전자이다.
조절 서열
본원에서 개시된 ASO 작용제는 치료적 유익성을 이끌어 낼 만큼 충분히 높은 수준으로 발현될 필요가 있을 수 있다. 따라서, 폴리뉴클레오티드 발현은 개시된 ASO 작용제의 견실한 발현을 주동할 수 있는 프로모터 서열에 의해 매개될 수 있다. 본원에서 개시된 방법과 조성물에 따라서, 프로모터는 이종성 프로모터일 수 있다. 본원에서 이용된 바와 같이, 용어 "이종성 프로모터"는 자연에서 소정의 인코딩 서열에 작동가능하게 연결되는 것으로 밝혀지지 않은 프로모터를 지칭한다. 유용한 이종성 제어 서열은 일반적으로, 포유류 또는 바이러스 유전자를 인코딩하는 서열로부터 유래된 것들을 포함한다.
본원 발명의 목적으로, 이종성 프로모터 및 다른 제어 요소 둘 모두, 예컨대 CNS 특이적 및 유도성 프로모터, 인핸서 등이 특히 유용할 것이다. 프로모터는 온전히 선천적 유전자 (예를 들면, Grik2 유전자)로부터 유래될 수 있거나 또는 상이한 자연 발생 프로모터로부터 유래된 상이한 요소로 구성될 수 있다. 대안으로, 프로모터는 합성 폴리뉴클레오티드 서열을 포함할 수 있다. 상이한 프로모터는 상이한 조직 또는 세포 유형에서, 또는 상이한 발달 단계에서, 또는 상이한 환경 조건 또는 약물 또는 전사 보조인자의 존재 또는 부재에 대한 반응으로 유전자의 발현을 주도할 것이다. 편재성, 세포 유형 특이적, 조직 특이적, 발달 단계 특이적 및 조건적 프로모터, 예를 들면, 약물 반응성 프로모터 (예를 들면 테트라사이클린 반응성 프로모터)는 당해 분야에서 널리 알려져 있다.
포유류 시스템에는, 3가지 종류의 프로모터가 존재하고 발현 벡터의 작제를 위한 후보이다: (i) 큰 리보솜 RNA의 전사를 제어하는 Pol I 프로모터; (ii) mRNA (이들은 단백질로 번역된다), 작은 핵 RNA (snRNA) 및 내인성 마이크로RNA (예를 들면, 프리-mRNA의 인트론으로부터)의 전사를 제어하는 Pol II 프로모터; 및 (iii) 작은 비코딩 RNA를 고유하게 전사하는 Pol III 프로모터. 각각은 생체내에서 RNA의 발현을 위한 작제물을 설계할 때 고려되는 이점 및 단점을 갖는다. 예를 들면, Pol III 프로모터는 생체내에서 DNA 주형으로부터 ASO 작용제 (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA, 또는 shmiRNA)를 합성하는 데 유용하다. 조직 특이적 발현에 대한 더 큰 제어를 위해, Pol II 프로모터가 선호되지만 단지 miRNA의 전사에만 이용될 수 있다. 하지만, Pol II 프로모터가 이용될 때, RNA가 siRNA, shRNA 또는 miRNA로서 기능하고 생체내에서 펩티드로 번역되지 않도록, 번역 개시 신호를 누락하는 것이 바람직할 수 있다.
본원에서 설명된 조성물과 방법용으로 적합한 폴리뉴클레오티드는 또한, 포유류 조절 서열, 예컨대 예를 들면, 프로모터 서열 및 임의적으로, 인핸서 서열의 제어하에, Grik2 mRNA를 표적으로 하는 ASO 작용제를 인코딩하는 것들을 포함한다. 포유류 세포에서 개시된 ASO 작용제의 발현에 유용한 예시적인 프로모터는 편재성 프로모터 예컨대 예를 들면, H1 프로모터, 7SK 프로모터, 아포지질단백질 E-인간-알파 1-항트립신 프로모터, CK8 프로모터, 뮤린 U1 프로모터 (mU1a), 신장 인자 1α (EF-1α) 프로모터, 티록신 결합 글로불린 (TBG) 프로모터, 포스포글리세르산염 키나아제 (PKG) 프로모터, CAG ((CMV) 시토메갈로바이러스 인핸서, 닭 베타 액틴 프로모터 (CBA) 및 토끼 베타 글로빈 인트론의 복합물), SV40 초기 프로모터, 뮤린 유방 종양 바이러스 LTR 프로모터; 아데노바이러스 주요 후기 프로모터 (Ad MLP); 단순 헤르페스 바이러스 (HSV) 프로모터, CMV 프로모터 예컨대 CMV 극초기 프로모터 영역 (CMV-IE), 라우스 육종 바이러스 (RSV) 프로모터, 그리고 U6 프로모터 또는 이의 변이체를 포함한다. 본원에서 개시된 저해성 RNA 서열의 세포 유형 특이적 발현을 주동하는 목적으로, 세포 유형 특이적 프로모터가 이용될 수 있다. 예를 들면, Grik2 ASO 작용제의 뉴런 특이적 발현은 뉴런 특이적 프로모터, 예컨대 예를 들면, 인간 시냅신 1 (hSyn) 프로모터, 헥사리보뉴클레오티드 결합 단백질-3 (NeuN) 프로모터, Ca2+/칼모듈린 의존성 단백질 키나아제 II (CaMKII) 프로모터, 튜불린 알파 I (Tα-1) 프로모터, 뉴런 특이적 에놀라아제 (NSE) 프로모터, 혈소판 유래 성장 인자 베타 사슬 (PDGFβ) 프로모터, 소포성 글루타민산염 전달체 (VGLUT) 프로모터, 소마토스타틴 (SST) 프로모터, 신경펩티드 Y (NPY) 프로모터, 혈관작용 장관 펩티드 (VIP) 프로모터, 파브알부민 (PV) 프로모터, 글루타민산염 탈카르복실화효소 (GAD65 또는 GAD67) 프로모터, 도파민-1 수용체 (DRD1) 및 도파민-2 수용체 (DRD2)의 프로모터, 미소관 관련 단백질 1B (MAP1B), 보체 성분 1 q 하위성분 유사 2 (C1ql2) 프로모터, 프로오피오멜라노코르틴 (POMC) 프로모터, 그리고 프로스페로 호메오복스 단백질 1 (PROX1) 프로모터를 이용하여 부여될 수 있다. hSyn 및 CaMKII 프로모터의 변이체가 Hioki et al. Gene Therapy 14:872-82 (2007) 및 Sauerwald et al. J. Biol. Chem. 265(25):14932-7 (1990)에서 이전에 기술되었는데, 이들의 개시는 이들이 특정한 hSyn 및 CaMKII 프로모터 서열에 관계하는 한에 있어서, 본원에서 참조로서 편입된다. 특이적으로 해마의 DG 세포에서 폴리뉴클레오티드 발현을 주동하는 데 적합한 프로모터는 C1ql2, POMC 및 PROX1 프로모터를 포함한다.
특정 실례에서, 본원 발명의 발현 벡터는 SYN 프로모터 (예를 들면, 예컨대 인간 SYN 프로모터 (hSyn), 예를 들면, 서열 번호: 682-685 및 790 중에서 하나 또는 서열 번호: 682-682 및 790 중에서 하나의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)를 포함한다. 다른 실례에서, 본원 발명의 발현 벡터는 CAMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나 또는 서열 번호: 687-691 및 802 중에서 하나의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)를 포함한다. 또 다른 실례에서, 본원 발명의 발현 벡터는 C1QL2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791 또는 서열 번호: 719 또는 서열 번호: 791의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)를 포함한다.
합성 프로모터, 하이브리드 프로모터 등이 또한, 본원에서 개시된 방법과 조성물용으로 이용될 수 있다. 이에 더하여, 비바이러스 유전자, 예컨대 뮤린 메탈로티오네인 유전자로부터 유래된 서열 역시 본원에서 용도를 발견할 것이다. 이런 프로모터 서열은 예를 들면, Stratagene (San Diego, CA)으로부터 상업적으로 가용하다. 발현 벡터 (예를 들면, 플라스미드 또는 바이러스 벡터, 예컨대 예를 들면, AAV 또는 렌티바이러스 벡터)용으로 적합한 예시적인 프로모터 서열은 아래의 표 5 및 표 6에서 제공된다.
표 5: 예시적인 뉴런 특이적 프로모터 서열
특정 실례에서, 본원 발명의 바이러스 벡터 (예를 들면, AAV 벡터)는 뉴런 특이적 프로모터 서열을 통합한다. 특정 실례에서, 뉴런 특이적 프로모터는 인간 Syn 프로모터, 예컨대, 서열 번호: 682-685 및 서열 번호: 790 중에서 하나의 핵산 서열, 또는 서열 번호: 682-685 및 서열 번호: 790 중에서 하나의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 인간 Syn 프로모터이다.
다른 실례에서, 뉴런 특이적 프로모터는 NeuN 프로모터 서열, 예컨대 서열 번호: 686 또는 서열 번호: 686의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체의 NeuN 프로모터 서열이다.
다른 실례에서, 뉴런 특이적 프로모터는 CaMKII 프로모터 서열, 예컨대 서열 번호: 687-691 및 서열 번호: 802 중에서 하나, 또는 서열 번호: 687-691 및 서열 번호: 802 중에서 하나의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체의 CaMKII 프로모터 서열이다.
추가의 CaMKII 프로모터는 Wang et al. (Mol. Biol. Rep. 35(1): 37-44, 2007)에서 설명된 인간 알파 CaMKII 프로모터 서열을 포함할 수 있는데, 이것의 개시는 CaMKII 프로모터 서열에 관계하는 한에 있어서, 본원에 온전히 통합된다.
다른 실례에서, 뉴런 특이적 프로모터는 NSE 프로모터 서열, 예컨대 서열 번호: 692 또는 서열 번호: 693, 또는 서열 번호: 692 또는 서열 번호: 693의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체의 NSE 프로모터 서열이다.
다른 실례에서, 뉴런 특이적 프로모터는 PDGFβ 프로모터 서열, 예컨대 서열 번호: 694-696 또는 서열 번호: 694-696의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체의 PDGFβ 프로모터 서열이다.
다른 실례에서, 뉴런 특이적 프로모터는 VGluT 프로모터 서열, 예컨대 서열 번호: 697-701, 또는 서열 번호: 708-712의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체의 VGluT 프로모터 서열이다.
다른 실례에서, 뉴런 특이적 프로모터는 SST 프로모터 서열, 예컨대 서열 번호: 702 또는 서열 번호: 703, 또는 서열 번호: 702 또는 서열 번호: 703의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체의 SST 프로모터 서열이다.
다른 실례에서, 뉴런 특이적 프로모터는 NPY 프로모터 서열, 예컨대 서열 번호: 704 또는 서열 번호: 704의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체의 NPY 프로모터 서열이다.
다른 실례에서, 뉴런 특이적 프로모터는 VIP 프로모터 서열, 예컨대 서열 번호: 705 또는 서열 번호: 706, 또는 서열 번호: 705 또는 서열 번호: 706의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체의 VIP 프로모터 서열이다.
다른 실례에서, 뉴런 특이적 프로모터는 PV 프로모터 서열, 예컨대 서열 번호: 707-709, 또는 서열 번호: 718-720의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체의 PV 프로모터 서열이다.
다른 실례에서, 뉴런 특이적 프로모터는 GAD65 프로모터 서열, 예컨대 서열 번호: 710-713, 또는 서열 번호: 710-713의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체의 GAD65 프로모터 서열이다.
다른 실례에서, 뉴런 특이적 프로모터는 GAD67 프로모터 서열, 예컨대 서열 번호: 714 또는 서열 번호: 715, 또는 서열 번호: 714 또는 서열 번호: 715의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체의 GAD67 프로모터 서열이다.
다른 실례에서, 뉴런 특이적 프로모터는 DRD1 프로모터 서열, 예컨대 서열 번호: 716 또는 서열 번호: 716의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체의 DRD1 프로모터 서열이다.
다른 실례에서, 뉴런 특이적 프로모터는 DRD2 프로모터 서열, 예컨대 서열 번호: 717 또는 서열 번호: 718, 또는 서열 번호: 717 또는 서열 번호: 718의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체의 DRD2 프로모터 서열이다.
다른 실례에서, 뉴런 특이적 프로모터는 C1ql2 프로모터 서열, 예컨대 서열 번호: 719, 또는 서열 번호: 719 또는 서열 번호: 791의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체의 C1ql2 프로모터 서열이다.
다른 실례에서, 뉴런 특이적 프로모터는 POMC 프로모터 서열, 예컨대 서열 번호: 720 또는 서열 번호: 720의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체의 POMC 프로모터 서열이다.
다른 실례에서, 뉴런 특이적 프로모터는 PROX1 프로모터 서열, 예컨대 서열 번호: 721 또는 서열 번호: 722, 또는 서열 번호: 737 또는 738의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체의 PROX1 프로모터 서열이다.
또 다른 실례에서, 뉴런 특이적 프로모터는 MAP1B 프로모터 서열, 예컨대 서열 번호: 723-725, 또는 서열 번호: 723-725의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체의 MAP1B 프로모터 서열이다.
또 다른 실례에서, 뉴런 특이적 프로모터는 Tα-1 프로모터 서열, 예컨대 서열 번호: 726 또는 서열 번호: 727, 또는 서열 번호: 726 또는 서열 번호: 727의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체의 Tα-1 프로모터 서열이다.
표 6: 예시적인 편재성 프로모터 서열
다른 실례에서, 본원 발명의 바이러스 벡터 (예를 들면, AAV 벡터)는 본원 발명의 안티센스 작제물을 발현할 수 있는 편재성 프로모터 서열을 통합한다. 한 가지 실례에서, 편재성 프로모터는 RNA Pol II 또는 RNA Pol III 프로모터이다. 예시적인 Pol II 및 Pol III 프로모터는 Preece et al. Gene Ther. 27:451-8(2020) 및 Jawdekar et al. Biochim. Biophys. Acta 1779(5):295-305 (2008)에서 설명되는데, 이들의 개시는 RNA Pol II 및 RNA Pol III 프로모터에 관계하는 한에 있어서, 본원에 참조로서 편입된다. 예를 들면, 본원 발명의 벡터 내로 포함하기 적합한 RNA Pol III 프로모터는 U6 작은 핵 1 프로모터, 예컨대, 서열 번호: 728-733 또는 772 중에서 하나의 핵산 서열, 또는 서열 번호: 728-733 또는 772 중에서 하나의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 U6 작은 핵 1 프로모터일 수 있다.
다른 실례에서, RNA Pol III 프로모터는 H1 프로모터, 예컨대 서열 번호: 734의 핵산 서열 또는 서열 번호: 734의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 H1 프로모터이다.
다른 실례에서, RNA Pol III 프로모터는 7SK 프로모터, 예컨대 서열 번호: 735의 핵산 서열 또는 서열 번호: 735의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 7SK 프로모터이다.
다른 실례에서, 편재성 프로모터는 아포지질단백질 E (ApoE)-인간 알파 1-항트립신 (hAAT; ApoE-hAAT) 프로모터, 예컨대 서열 번호: 736의 핵산 서열 또는 서열 번호 736의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 ApoE-hAAT 프로모터이다.
다른 실례에서, 편재성 프로모터는 시토메갈로바이러스 (CMV) 초기 인핸서 요소, 닭 베타-액틴 유전자의 프로모터, 첫 번째 엑손 및 첫 번째 인트론, 그리고 토끼 베타-글로빈 유전자의 스플라이스 수용자를 포함하는 CAG 프로모터, 예컨대 서열 번호: 737의 핵산 서열 또는 서열 번호 737의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 CAG 프로모터이다.
다른 실례에서, 편재성 프로모터는 닭 베타 액틴 (CBA) 프로모터, 예컨대 서열 번호: 738의 핵산 서열 또는 서열 번호: 738의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 CBA 프로모터이다.
다른 실례에서, 편재성 프로모터는 근육 크레아틴 키나아제 프로모터의 변이체인 CK8 프로모터, 예컨대 서열 번호: 739의 핵산 서열 또는 서열 번호: 739의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 CK8 프로모터이다.
다른 실례에서, 편재성 프로모터는 생쥐 U1 작은 핵 RNA (mU1a) 프로모터, 예컨대 서열 번호: 740의 핵산 서열 또는 서열 번호: 740의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 mU1a 프로모터이다.
다른 실례에서, 편재성 프로모터는 신장 인자 1 알파 (EF-1α) 프로모터, 예컨대 서열 번호: 741의 핵산 서열 또는 서열 번호: 741의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 EF-1α 프로모터이다.
다른 실례에서, 편재성 프로모터는 티록신 결합 글로불린 (TBG) 프로모터, 예컨대 서열 번호: 742의 핵산 서열 또는 서열 번호: 742의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 TBG 프로모터이다.
일단 개시된 ASO 작용제를 인코딩하는 폴리뉴클레오티드가 포유류 세포의 핵 DNA 내로 통합되면, 이러한 폴리뉴클레오티드의 전사는 당해 분야에서 공지된 방법에 의해 유도될 수 있다. 예를 들면, 포유류 세포를 외부 화학 시약, 예컨대 포유류 프로모터에 대한 전사 인자 및/또는 RNA 중합효소의 결합을 조정하여 유전자 발현을 조절하는 작용제에 노출시킴으로써 발현이 유도될 수 있다. 화학 시약은 예를 들면, 포유류 프로모터에 결합한 억제인자 단백질을 제거함으로써, 상기 프로모터에 대한 RNA 중합효소 및/또는 전사 인자의 결합을 가능하게 하는 데 역할을 할 수 있다. 대안으로, 화학 시약은 포유류 프로모터의 하류에 위치된 유전자의 전사 속도가 이러한 화학 시약의 존재에서 증가되도록, RNA 중합효소 및/또는 전사 인자에 대한 상기 프로모터의 친화성을 증강하는 데 역할을 할 수 있다. 상기 기전에 의해 폴리뉴클레오티드 전사를 강력하게 만드는 화학 시약의 실례는 테트라사이클린 및 독시사이클린이다. 이들 시약은 상업적으로 가용하고 (Life Technologies, Carlsbad, CA), 그리고 확립된 프로토콜에 따라서 유전자 발현을 증진하기 위해 포유류 세포에 투여될 수 있다.
본원에서 설명된 조성물과 방법에 이용하기 위한 폴리뉴클레오티드 내에 포함될 수 있는 다른 DNA 서열 요소는 인핸서 서열이다. 인핸서는 DNA가 전사 개시 부위에서 전사 인자 및 RNA 중합효소의 결합에 대해 호의적인 3차원 방향을 채택하도록, 관심되는 유전자를 내포하는 폴리뉴클레오티드에서 입체형태적 변화를 유도하는 조절 요소의 다른 부류를 나타낸다. 따라서, 본원에서 설명된 조성물과 방법에 이용하기 위한 폴리뉴클레오티드는 Grik2 표적화 ASO 작용제를 인코딩하고 포유류 인핸서 서열을 부가적으로 포함하는 것들을 포함한다. 포유류 유전자로부터 유래된 많은 인핸서 서열이 현재 알려져 있고, 그리고 실례는 포유류 글로빈, 엘라스타아제, 알부민, α-태아단백질 및 인슐린을 인코딩하는 유전자로부터 유래된 인핸서이다. 본원에서 설명된 조성물과 방법에 이용하기 위한 인핸서는 또한, 진핵 세포를 감염시킬 수 있는 바이러스의 유전 물질로부터 유래되는 것들을 포함한다. 실례는 복제 기점의 후기 측면에서 SV40 인핸서 (bp 100-270), 시토메갈로바이러스 초기 프로모터 인핸서, 복제 기점의 후기 측면에서 폴리오마 인핸서, 그리고 아데노바이러스 인핸서이다. 진핵 유전자 전사의 활성화를 유도하는 추가의 인핸서 서열은 Yaniv et al., Nature 297:17 (1982)에서 개시된다. 인핸서는 예를 들면, 이러한 유전자의 5' 또는 3' 위치에서 본원 발명의 안티센스 작제물을 인코딩하는 폴리뉴클레오티드를 내포하는 벡터 내로 스플라이싱될 수 있다. 특정 배향에서, 인핸서는 프로모터의 5' 측면에 배치되고, 상기 프로모터는 차례로 본원 발명의 ASO 작용제를 인코딩하는 폴리뉴클레오티드에 비하여 5'에 위치된다. 인핸서 서열의 무제한적 실례는 아래의 표 7에서 제공된다.
본원에서 설명된 조성물과 방법에 이용하기 위한 폴리뉴클레오티드 내에 포함될 수 있는 추가의 조절 요소는 인트론 서열이다. 인트론 서열은 성숙 mRNA 산물을 생산하기 위해 RNA 스플라이싱 동안 제거되는 프리-mRNA에서 발견되는 비단백질-코딩 RNA 서열이다. 인트론 서열은 이들이 다른 비코딩 RNA 분자를 생산하기 위해 더욱 처리될 수 있다는 점에서 유전자 발현의 조절에 중요하다. 대안적 스플라이싱, 넌센스 매개 붕괴, 그리고 mRNA 이출이 인트론 서열에 의해 조절되는 것으로 밝혀진 생물학적 과정이다. 인트론 서열은 또한, 인트론 매개 증강을 통해 도입유전자의 발현을 가능하게 할 수 있다. 인트론 서열의 무제한적 실례는 아래의 표 7에서 제공된다.
본원 발명의 벡터와 함께 이용될 수 있는 추가 조절 요소는 반전 말단 반복 (ITR) 서열을 포함한다. ITR 서열은 예를 들면 AAV 유전체의 5'와 3' 단부에서 발견되고, 각각 약 145개의 염기쌍을 전형적으로 내포한다. AAV ITR 서열은 일단 AAV 벡터가 세포 내로 통합되면, 상보성 가닥 합성을 가능하게 함으로써 AAV 유전체 증식에 특히 중요하다. 게다가, ITR은 숙주 세포의 유전체 내로 AAV 유전체의 통합 및 AAV 유전체의 캡시드화에 결정적인 것으로 밝혀졌다. ITR 서열의 무제한적 실례는 아래의 표 7에서 제공된다.
본원 발명의 벡터 내로 통합에 적합한 추가의 조절 요소는 폴리아데닐화 서열 (다시 말하면, 폴리A 서열)을 포함한다. 폴리A 서열은 아데닌 염기의 스트레치를 내포하는 RNA 꼬리이다. 이들 서열은 성숙 mRNA 전사체를 생산하기 위해 RNA 분자의 3' 단부에 부가된다. 핵 이출, 번역 및 안정성을 비롯한, mRNA 처리와 수송에 관련된 여러 생물학적 과정이 폴리A 서열에 의해 조정된다. 포유류 세포에서, 폴리A 꼬리의 단축은 mRNA 분해의 가능성을 증가시켰다. 폴리A 서열의 무제한적 실례는 아래의 표 7에서 제공된다.
표 7: 예시적인 조절 서열
다른 실례에서, 본원 발명의 바이러스 벡터 (예를 들면, AAV 벡터)는 본원 발명의 안티센스 작제물의 발현을 가능하게 할 수 있는 하나 이상의 조절 서열 요소를 통합한다. 한 가지 실례에서, 조절 서열 요소는 인트론 서열이다. 예를 들면, 본원 발명의 벡터 내로 포함하기 적합한 인트론 서열은 키메라 인트론, 예컨대 서열 번호: 743의 핵산 서열 또는 서열 번호: 743의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 키메라 인트론일 수 있다.
다른 실례에서, 인트론 서열은 면역글로불린 중쇄-가변 4 (VH4) 인트론, 예컨대 서열 번호: 744의 핵산 서열 또는 서열 번호: 744의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 VH4 서열이다.
다른 실례에서, 조절 서열 요소는 인핸서 서열이다. 예를 들면, 인핸서 서열은 CMV 인핸서, 예컨대 서열 번호: 745의 핵산 서열 또는 서열 번호: 745의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 CMV 인핸서일 수 있다.
다른 실례에서, 조절 서열 요소는 ITR 서열, 예컨대 예를 들면, AAV ITR 서열이다. 예를 들면, ITR 서열은 AAV 5' ITR 서열, 예컨대 서열 번호: 746 또는 서열 번호: 747의 핵산 서열, 또는 서열 번호: 746 또는 서열 번호: 747의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 AAV 5' ITR 서열일 수 있다.
다른 실례에서, ITR 서열은 AAV 3' ITR 서열, 예컨대 서열 번호: 748 또는 서열 번호: 749의 핵산 서열, 또는 서열 번호: 748 또는 서열 번호: 749의 핵산 서열에 적어도 70% (예를 들면, 적어도 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 AAV 3' ITR 서열이다.
다른 실례에서, 조절 서열 요소는 폴리아데닐화 신호 (다시 말하면, 폴리A 꼬리)이다. 예를 들면, 본원에서 개시된 벡터용으로 적합한 폴리아데닐화 신호는 토끼 β-글로빈 (RBG) 폴리아데닐화 신호, 예컨대 서열 번호: 750, 서열 번호: 751 또는 서열 번호: 792의 핵산 서열, 또는 서열 번호: 750, 서열 번호: 751 또는 서열 번호: 792의 핵산 서열에 적어도 70% (예를 들면, 적어도 71%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 RBG 폴리아데닐화 신호를 포함한다. 개시된 조성물 및 방법과 함께 이용될 수 있는 다른 폴리아데닐화 신호는 소 성장 호르몬 (BGH) 폴리아데닐화 신호, 예컨대 서열 번호: 793 또는 서열 번호: 793의 핵산 서열에 적어도 70% (예를 들면, 적어도 71%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 그 초과)의 서열 동일성을 갖는 이의 변이체의 BGH 폴리아데닐화 신호이다.
바이러스 벡터
바이러스 유전체는 포유류 세포 내로 외인성 폴리뉴클레오티드의 효율적인 전달에 이용될 수 있는 벡터의 풍부한 공급원을 제공한다. 바이러스 유전체는 유전자 전달에 특히 유용한 벡터인데, 그 이유는 이런 유전체 내에 내포된 폴리뉴클레오티드가 전형적으로, 일반적인 또는 특수한 형질도입에 의해 포유류 세포의 핵 유전체 내로 통합되기 때문이다. 이들 과정은 자연 바이러스 복제 주기의 일부로서 발생하고, 그리고 유전자 통합을 유도하기 위한 추가의 단백질 또는 시약을 필요로 하지 않는다. 바이러스 벡터의 실례는 파보바이러스 (예를 들면, 아데노 관련 바이러스 (AAV)), 레트로바이러스 (예를 들면, 레트로바이러스과 (Retroviridae) 바이러스 벡터), 아데노바이러스 (예를 들면, Ad5, Ad26, Ad34, Ad35 및 Ad48), 코로나바이러스, 음성 가닥 RNA 바이러스 예컨대 오르토믹소바이러스 (예를 들면, 인플루엔자 바이러스), 랍도바이러스 (예를 들면, 광견병 및 수포성 구내염 바이러스), 파라믹소바이러스 (예를 들면 홍역 및 센다이), 양성 가닥 RNA 바이러스, 예컨대 피코르나바이러스 및 알파바이러스, 그리고 아데노바이러스, 헤르페스바이러스 (예를 들면, 단순 헤르페스 바이러스 1형과 2형, 엡스타인 바르 바이러스, 시토메갈로바이러스) 및 폭스바이러스 (예를 들면, 우두, 변형된 우두 앙카라 (MVA), 계두 및 카나리아 두창)을 비롯한 이중 가닥 DNA 바이러스이다. 다른 바이러스는 예를 들면, 노워크 바이러스, 토가바이러스, 플라비바이러스, 레오바이러스, 파포바바이러스, 헤파드나바이러스, 인간 유두종 바이러스, 인간 거품형성 바이러스, 그리고 간염 바이러스를 포함한다. 레트로바이러스의 실례는 조류 백혈증-육종, 조류 C-형 바이러스, 포유류 C-형, B-형 바이러스, D-형 바이러스, 온코레트로바이러스, HTLV-BLV 군, 렌티바이러스, 알파레트로바이러스, 감마레트로바이러스, 스푸마바이러스이다 (Coffin, J. M., Retroviridae: The viruses and their replication, Virology, Third Edition (Lippincott-Raven, Philadelphia, (1996))). 다른 실례는 뮤린 백혈병 바이러스, 뮤린 육종 바이러스, 뮤린 유방 종양 바이러스, 소 백혈병 바이러스, 고양이 백혈병 바이러스, 고양이 육종 바이러스, 조류 백혈병 바이러스, 인간 T 세포 백혈병 바이러스, 개코원숭이 내인성 바이러스, 긴팔 원숭이 백혈병 바이러스, 메이슨 화이자 원숭이 바이러스, 유인원 면역결핍 바이러스, 유인원 육종 바이러스, 라우스 육종 바이러스 및 렌티바이러스이다. 벡터의 다른 실례는 예를 들면 McVey et al., (U.S. 특허 번호 5,801,030)에서 설명되는데, 이것의 교시는 본원에서 참조로서 편입된다.
AAV 벡터
본원에서 설명된 조성물의 핵산은 예를 들면, 본원에서 개시된 방법과 관련하여 그들의 세포 내로의 도입을 용이하게 하기 위해 AAV 벡터 및/또는 AAV 비리온 내로 통합될 수 있다. AAV 벡터는 중추신경계에서 이용될 수 있고, 그리고 적합한 프로모터 및 혈청형은 예를 들면, Pignataro et al., J Neural Transm 125(3):575-89 (2017)에서 논의되는데, 이것의 개시는 이것이 CNS 유전자 요법에서 유용한 프로모터 및 AAV 혈청형과 관련되는 한에 있어서, 본원에서 참조로서 편입된다. 본원에서 설명된 조성물과 방법에서 유용한 rAAV 벡터는 (1) 발현되는 이종성 서열 (예를 들면, Grik2 mRNA 표적화 ASO 작용제를 인코딩하는 폴리뉴클레오티드) 및 (2) 이종성 유전자의 통합과 발현을 가능하게 하는 바이러스 서열을 포함하는 재조합 핵산 작제물이다. 바이러스 서열은 DNA의 복제 및 비리온 내로의 포장을 위해 시스로 필요한, AAV의 그런 서열 (예를 들면, 기능적 ITR)을 포함할 수 있다. 이런 rAAV 벡터는 또한, 마커 또는 리포터 유전자를 내포할 수 있다. 유용한 rAAV 벡터는 AAV WT 유전자 중에서 하나 이상이 전체적으로 또는 부분적으로 결실되지만, 기능적 측접 ITR 서열을 유지한다. AAV ITR은 특정 적용에 적합한 임의의 혈청형일 수 있다. rAAV 벡터를 이용하기 위한 방법은 예를 들면, Tai et al., J. Biomed. Sci. 7:279 (2000) 및 Monahan and Samulski, Gene Delivery 7:24 (2000)에서 설명되는데, 이들 각각의 개시는 이들이 유전자 전달을 위한 AAV 벡터와 관련되는 한에 있어서, 본원에서 참조로서 편입된다.
본원 발명의 ASO 작용제 (예를 들면, 본원에서 설명된 siRNA, shRNA, miRNA, 또는 shmiRNA, 또는 shmiRNA)를 통합하기 위한 벡터로서 이용될 수 있는 AAV의 실례는 예를 들면, AAV1, AAV2, AAV3, AAV4, AAV5, AAV6, AAV7, AAV8, AAV9, AAV10, AAV11, AAV12, AAV13, AAV14, AAV15, AAV16, AAV.rh8, AAV.rh10, AAV.rh20, AAV.rh39, AAV.Rh74, AAV.RHM4-1, AAV.hu37, AAV.Anc80, AAV.Anc80L65, AAV.7m8, AAV.PHP.B, AAV.PHP.EB, AAV2.5, AAV2tYF, AAV3B, AAV.LK03, AAV.HSC1, AAV.HSC2, AAV.HSC3, AAV.HSC4, AAV.HSC5, AAV.HSC6, AAV.HSC7, AAV.HSC8, AAV.HSC9, AAV.HSC10, AAV.HSC11, AAV.HSC12, AAV.HSC13, AAV.HSC14, AAV.HSC15, AAV-TT, AAV-DJ8, 그리고 AAV.HSC16을 포함한다.
본원에서 설명된 핵산 및 벡터는 세포 내로 핵산 또는 벡터의 도입을 가능하게 하기 위해 rAAV 비리온 내로 통합될 수 있다. AAV의 캡시드 단백질은 비리온의 외부, 비-핵산 부분을 구성하고 AAV cap 유전자에 의해 인코딩된다. cap 유전자는 비리온 조립에 필요한 3가지 바이러스 외피 단백질, VP1, VP2 및 VP3을 인코딩한다. rAAV 비리온의 작제는 예를 들면 US 5,173,414; US 5,139,941; US 5,863,541; US 5,869,305; US 6,057,152; 및 US 6,376,237에서뿐만 아니라 Rabinowitz et al., J. Virol. 76:791 (2002) 및 Bowles et al., J. Virol. 77:423 (2003)에서 설명되었는데, 이들 각각의 개시는 이들이 유전자 전달을 위한 AAV 벡터와 관련되는 한에 있어서, 본원에서 참조로서 편입된다.
본원에서 설명된 조성물과 방법용으로 유용한 rAAV 비리온은 AAV 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 및 rh74를 비롯한 다양한 AAV 혈청형으로부터 유래된 것들을 포함한다. 중추신경계 내에 위치되거나 또는 이것으로 전달된 세포를 표적으로 하기 위해, AAV2, AAV9 및 AAV10이 특히 유용할 수 있다. 상이한 혈청형의 AAV 벡터 및 AAV 단백질의 작제와 이용은 예를 들면 Chao et al., Mol. Ther. 2:619 (2000); Davidson et al., Proc. Natl. Acad. Sci. USA 97:3428 (2000); Xiao et al., J. Virol. 72:2224 (1998); Halbert et al., J. Virol. 74:1524 (2000); Halbert et al., J. Virol. 75:6615 (2001); 및 Auricchio et al., Hum. Molec. Genet. 10:3075 (2001)에서 설명되는데, 이들 각각의 개시는 이들이 유전자 전달을 위한 AAV 벡터와 관련되는 한에 있어서, 본원에서 참조로서 편입된다.
위유형화 rAAV 벡터가 또한, 본원에서 설명된 조성물과 방법용으로 유용하다. 위유형화 벡터는 소정의 혈청형 (AAV1, AAV2, AAV3, AAV4, AAV5, AAV6, AAV7, AAV8, AAV9, AAV10, AAV11, AAV12, AAV13, AAV14, AAV15, AAV16, AAV.rh8, AAV.rh10, AAV.rh20, AAV.rh39, AAV.Rh74, AAV.RHM4-1, AAV.hu37, AAV.Anc80, AAV.Anc80L65, AAV.7m8, AAV.PHP.B, AAV.PHP.EB, AAV2.5, AAV2tYF, AAV3B, AAV.LK03, AAV.HSC1, AAV.HSC2, AAV.HSC3, AAV.HSC4, AAV.HSC5, AAV.HSC6, AAV.HSC7, AAV.HSC8, AAV.HSC9, AAV.HSC10, AAV.HSC11, AAV.HSC12, AAV.HSC13, AAV.HSC14, AAV.HSC15, AAV-TT, AAV-DJ8, 또는 AAV.HSC16) 이외의 혈청형으로부터 유래된 캡시드 유전자로 위유형화된 소정의 혈청형의 AAV 벡터를 포함한다. 예를 들면, AAV는 위유형화 재조합 AAV (rAAV) 벡터, 예컨대 예를 들면, rAAV2/8 또는 rAAV2/9 벡터를 포함할 수 있다. 위유형화 rAAV를 생산하고 이용하기 위한 방법은 당해 분야에서 알려져 있다 (참조: 예를 들면, Duan et al., J. Virol., 75:7662-7671 (2001); Halbert et al., J. Virol., 74:1524-1532 (2000); Zolotukhin et al., Methods 28:158-167 (2002); 및 Auricchio et al., Hum. Molec. Genet. 10:3075-3081, (2001).
비리온 캡시드 내에 돌연변이를 갖는 AAV 비리온은 비돌연변이된 캡시드 비리온보다 효과적으로, 특정 세포 유형을 감염시키는 데 이용될 수 있다. 예를 들면, 적합한 AAV 돌연변이체는 AAV의 특정한 세포 유형으로의 표적화의 촉진을 위한 리간드 삽입 돌연변이를 가질 수 있다. 삽입 돌연변이체, 알라닌 선별검사 돌연변이체 및 에피토프 태그 돌연변이체를 비롯한 AAV 캡시드 돌연변이체의 작제와 특징화는 Wu et al., J. Virol. 74:8635 (2000)에서 설명된다. 본원에서 설명된 방법에서 이용될 수 있는 다른 rAAV 비리온은 바이러스의 분자 육종에 의해서뿐만 아니라 엑손 셔플링에 의해 생성되는 그런 캡시드 하이브리드를 포함한다. 예를 들면, Soong et al., Nat. Genet., 25:436 (2000) 및 Kolman and Stemmer, Nat. Biotechnol. 19:423 (2001)을 참조한다.
본원 발명의 조성물과 방법에서 이용되는 rAAV는 AAV1, AAV2, AAV3, AAV4, AAV5, AAV6, AAV7, AAV8, AAV9, AAV10, AAV11, AAV12, AAV13, AAV14, AAV15, AAV16, AAV.rh8, AAV.rh10, AAV.rh20, AAV.rh39, AAV.Rh74, AAV.RHM4-1, AAV.hu37, AAV.Anc80, AAV.Anc80L65, AAV.7m8, AAV.PHP.B, AAV.PHP.EB, AAV2.5, AAV2tYF, AAV3B, AAV.LK03, AAV.HSC1, AAV.HSC2, AAV.HSC3, AAV.HSC4, AAV.HSC5, AAV.HSC6, AAV.HSC7, AAV.HSC8, AAV.HSC9, AAV.HSC10, AAV.HSC11, AAV.HSC12, AAV.HSC13, AAV.HSC14, AAV.HSC15, AAV.HSC16, AAV-TT, AAVDJ8, 또는 이의 유도체, 변형 또는 위유형에서 선택되는 AAV 캡시드 혈청형으로부터 유래된 캡시드 단백질, 예컨대 예를 들면, AAV1, AAV2, AAV3, AAV4, AAV5, AAV6, AAV7, AAV8, AAV9, AAV10, AAV11, AAV12, AAV13, AAV14, AAV15, AAV16, AAV.rh8, AAV.rh10, AAV.rh20, AAV.rh39, AAV.Rh74, AAV.RHM4-1, AAV.hu37, AAV.Anc80, AAV.Anc80L65, AAV.7m8, AAV.PHP.B, AAV.PHP.EB, AAV2.5, AAV2tYF, AAV3B, AAV.LK03, AAV.HSC1, AAV.HSC2, AAV.HSC3, AAV.HSC4, AAV.HSC5, AAV.HSC6, AAV.HSC7, AAV.HSC8, AAV.HSC9, AAV.HSC10, AAV.HSC11, AAV.HSC12, AAV.HSC13, AAV.HSC14, AAV.HSC15, AAV-TT, AAV-DJ8 또는 AAV.HSC16에서 선택되는 AAV 캡시드 혈청형의 vp1, vp2 및/또는 vp3 서열과 예를 들면, 적어도 80% 또는 그 초과로 동일한, 예를 들면, 85%, 85%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99.5%, 또는 그 초과, 다시 말하면, 100%까지 동일한 캡시드 단백질을 포함할 수 있다.
본원에서 설명된 방법에서 이용될 수 있는 AAV 벡터는, 온전히 참조로서 편입되는 Zinn et al., 2015: 1056-1068에서 설명된 바와 같이, Anc80 또는 Anc80L65 벡터일 수 있다. AAV 벡터는, 본원에서 각각 온전히 참조로서 편입되는 미국 특허 번호 9,193,956; 9458517; 9,587,282 및 US 특허 출원 공개 번호 2016/0376323에서 설명된 바와 같이, 다음의 아미노산 삽입: LGETTRP ('956, '517, '282, 또는 '323의 서열 번호: 14) 또는 LALGETTRP ('956, '517, '282, 또는 '323의 서열 번호: 15) 중에서 하나를 포함할 수 있다. 대안으로, 본원에서 설명된 방법에서 이용되는 AAV 벡터는, 본원에서 각각 온전히 참조로서 편입되는 미국 특허 번호 9,193,956; 9,458,517; 9,587,282 및 US 특허 출원 공개 번호 2016/0376323에서 설명된 바와 같은 AAV.7m8일 수 있다. 더 나아가, 본원에서 설명된 방법에서 이용되는 AAV 벡터는 미국 특허 번호 9,585,971에서 개시된 임의의 AAV, 예컨대 AAV-PHP.B 벡터일 수 있다. 본원에서 설명된 방법에서 이용되는 다른 AAV 벡터는 Chan et al. (Nat Neurosci. 20(8):1172-1179, 2017)에서 개시된 임의의 벡터, 예컨대 아미노산 위치 588 및 589 사이에 삽입된 펩티드 및 변형 A587D/588G를 갖는 AAV9 캡시드 단백질을 포함하는 AAV.PHP.eB일 수 있다. 게다가, 본원에서 설명된 방법에서 이용되는 AAV 벡터는, 본원에서 각각 온전히 참조로서 편입되는 미국 특허 번호 9,840,719 및 WO 2015/013313에서 개시된 임의의 AAV, 예컨대 AAV.Rh74 또는 RHM4-1 벡터일 수 있다. 추가적으로, 본원에서 설명된 방법에서 이용되는 AAV 벡터는, 본원에서 온전히 참조로서 편입되는 WO 2014/172669에서 개시된 임의의 AAV, 예컨대 AAV rh.74일 수 있다. 본원에서 설명된 방법에서 이용되는 AAV 벡터는 또한, 각각 온전히 참조로서 편입되는 Georgiadis et al., 2016, Gene Therapy 23: 857-862 및 Georgiadis et al., 2018, Gene Therapy 25: 450에서 설명된 바와 같은 AAV2/5 벡터일 수 있다. 추가 실례에서, 본원에서 설명된 방법에서 이용되는 AAV 벡터는, 본원에서 온전히 참조로서 편입되는 WO 2017/070491에서 개시된 임의의 AAV, 예컨대 AAV2tYF 벡터일 수 있다. 추가적으로, 본원에서 설명된 방법에서 이용되는 AAV 벡터는, 온전히 참조로서 편입되는 Puzzo et al., 2017, Sci. Transl. Med. 29(9): 418에서 설명된 바와 같은 AAVLK03 또는 AAV3B 벡터일 수 있다. 추가 실례에서, 본원에서 설명된 방법에서 이용되는 AAV 벡터는, 각각 온전히 참조로서 편입되는 US 특허 번호 8,628,966; US 8,927,514; US 9,923,120 및 WO 2016/049230에서 개시된 임의의 AAV, 예컨대 HSC1, HSC2, HSC3, HSC4, HSC5, HSC6, HSC7, HSC8, HSC9, HSC10, HSC11, HSC12, HSC13, HSC14, HSC15, 또는 HSC16일 수 있다.
게다가, 본원에서 설명된 방법에서 이용되는 AAV 벡터는, 본원에서 각각 온전히 참조로서 편입되는 임의의 하기 특허와 특허 출원: 미국 특허 번호 7,282,199; 7,906,111; 8,524,446; 8,999,678; 8,628,966; 8,927,514; 8,734,809; US 9,284,357; 9,409,953; 9,169,299; 9,193,956; 9458517; 및 9,587,282; US 특허 출원 공개 번호 2015/0374803; 2015/0126588; 2017/0067908; 2013/0224836; 2016/0215024; 2017/0051257; 그리고 국제 특허 출원 번호 PCT/US2015/034799; PCT/EP2015/053335에서 개시된 AAV 벡터일 수 있다. rAAV 벡터는, 본원에서 각각 온전히 참조로서 편입되는 임의의 하기 특허와 특허 출원: 미국 특허 번호 7,282,199; 7,906,111; 8,524,446; 8,999,678; 8,628,966; 8,927,514; 8,734,809; US 9,284,357; 9,409,953; 9,169,299; 9,193,956; 9458517; 및 9,587,282; US 특허 출원 공개 번호 2015/0374803; 2015/0126588; 2017/0067908; 2013/0224836; 2016/0215024; 2017/0051257; 그리고 국제 특허 출원 번호 PCT/US2015/034799; PCT/EP2015/053335에서 개시된 AAV 캡시드의 vp1, vp2 및/또는 vp3 아미노산 서열에 적어도 80% 또는 그 초과로 동일한, 예를 들면, 85%, 85%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99.5%, 또는 그 초과로 동일한 캡시드 단백질을 가질 수 있다.
추가적으로, rAAV 벡터는, 각각의 내용이 본원에서 온전히 참조로서 편입되는 국제 출원 공개 번호 WO 2003/052051 (참조: 예를 들면, '051의 서열 번호: 2), WO 2005/033321 (참조: 예를 들면, '321의 서열 번호: 123 및 88), WO 03/042397 (참조: 예를 들면, '397의 서열 번호: 2, 81, 85 및 97), WO 2006/068888 (참조: 예를 들면, '888의 서열 번호: 1 및 3-6), WO 2006/110689 (참조: 예를 들면, '689의 서열 번호: 5-38), WO2009/104964 (참조: 예를 들면, '964의 서열 번호: 1-5, 7, 9, 20, 22, 24 및 31), W0 2010/127097 (참조: 예를 들면, '097의 서열 번호: 5-38), WO 2015/191508 (참조: 예를 들면, '058의 서열 번호: 80-294), 그리고 U.S. 출원 공개 번호 20150023924 (참조: 예를 들면, '924의 서열 번호: 1, 5-10)에서 개시된 캡시드 단백질, 예컨대 예를 들면, 국제 출원 공개 번호 WO 2003/052051 (참조: 예를 들면, '051의 서열 번호: 2), WO 2005/033321 (참조: 예를 들면, '321의 서열 번호: 123 및 88), WO 03/042397 (참조: 예를 들면, '397의 서열 번호: 2, 81, 85 및 97), WO 2006/068888 (참조: 예를 들면, '888의 서열 번호: 1 및 3-6), WO 2006/110689 (참조: 예를 들면, '689의 서열 번호: 5-38), WO2009/104964 (참조: 예를 들면, '964의 서열 번호: 1-5, 7, 9, 20, 22, 24 및 31), W0 2010/127097 (참조: 예를 들면, '097의 서열 번호: 5-38), WO 2015/191508 (참조: 예를 들면, '058의 서열 번호: 80-294), 그리고 U.S. 출원 공개 번호 20150023924 (참조: 예를 들면, '924의 서열 번호: 1, 5-10)에서 개시된 AAV 캡시드의 vp1, vp2 및/또는 vp3 아미노산 서열에 적어도 80% 또는 그 초과로 동일한, 예를 들면, 85%, 85%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99.5%, 또는 그 초과로 동일한 캡시드 단백질을 갖는 rAAV 벡터일 수 있다.
AAV 기반 바이러스 벡터의 핵산 서열 및 재조합 AAV와 AAV 캡시드를 만드는 방법은 예를 들면, 미국 특허 번호 7,282,199; 7,906,111; 8,524,446; 8,999,678; 8,628,966; 8,927,514; 8,734,809; US 9,284,357; 9,409,953; 9,169,299; 9,193,956; 9458517; 및 9,587,282; US 특허 출원 공개 번호 2015/0374803; 2015/0126588; 2017/0067908; 2013/0224836; 2016/0215024; 2017/0051257; 국제 특허 출원 번호 PCT/US2015/034799; PCT/EP2015/053335; WO 2003/052051, WO 2005/033321, WO 03/042397, WO 2006/068888, WO 2006/110689, WO2009/104964, W0 2010/127097 및 WO 2015/191508, 그리고 U.S. 출원 공개 번호 20150023924에서 교시된다.
따라서, rAAV 벡터는 2가지 이상의 AAV 캡시드 혈청형, 예컨대 예를 들면, AAV1, AAV2, AAV3, AAV4, AAV5, AAV6, AAV7, AAV8, AAV9, AAV10, AAV11, AAV12, AAV13, AAV14, AAV15, AAV16, AAV.rh8, AAV.rh10, AAV.rh20, AAV.rh39, AAV.Rh74, AAV.RHM4-1, AAV.hu37, AAV.Anc80, AAV.Anc80L65, AAV.7m8, AAV.PHP.B, AAV.PHP.EB, AAV2.5, AAV2tYF, AAV3B, AAV.LK03, AAV.HSC1, AAV.HSC2, AAV.HSC3, AAV.HSC4, AAV.HSC5, AAV.HSC6, AAV.HSC7, AAV.HSC8, AAV.HSC9, AAV.HSC10, AAV.HSC11, AAV.HSC12, AAV.HSC13, AAV.HSC14, AAV.HSC15, AAV-TT, AAV-DJ8, 또는 AAV.HSC16에서 선택되는 AAV 혈청형으로부터 유래된 캡시드 단백질을 내포하는 캡시드를 포함할 수 있다.
단일 가닥 AAV (ssAAV) 벡터가 개시된 방법과 조성물용으로 이용될 수 있다. 대안으로, 자가 상보성 AAV 벡터 (scAAV)가 이용될 수 있다 (참조: 예를 들면, Wu, 2007, Human Gene Therapy, 18(2):171-82, McCarty et al, 2001, Gene Therapy, Vol. 8, Number 16, Pages 1248-1254; 및 U.S. 특허 번호 6,596,535; 7,125,717; 및 7,456,683, 이들은 각각 본원에서 온전히 참조로서 편입된다).
뉴런 및/또는 신경아교 세포를 포함하지만 이들에 한정되지 않는, 중추신경계의 세포에 대한 향성을 갖는 재조합 AAV 벡터가 본원 발명의 폴리뉴클레오티드 작용제 (예를 들면, ASO 작용제)를 전달하는 데 이용될 수 있다. 이런 벡터는 비복제성 "rAAV" 벡터, 특히 AAV9 또는 AAVrh10 캡시드를 보유하는 것들을 포함할 수 있다. 본원에서 온전히 참조로서 편입되는 US 특허 번호 7,906,111에서 Wilson에 의해 설명된 것들 (AAV/hu.31 및 AAV/hu.32이 특히 선호된다)뿐만 아니라 본원에서 각각 온전히 참조로서 편입되는 US 특허 번호 8,628,966, US 특허 번호 8,927,514 및 Smith et al., 2014, Mol Ther 22: 1625-1634에서 Chatterjee에 의해 설명된 AAV 변이체 캡시드를 포함하지만 이들에 한정되지 않는 AAV 변이체 캡시드가 이용될 수 있다. 게다가, 자연 AAV2 분리주 사이에서 보존되는 아미노산 서열을 통합하는, Tordo et al. (Brain 141:2014-31, 2018; 본원에서 온전히 참조로서 편입됨)에 의해 개시된 AAV-TT 벡터 또한, 본원 발명의 조성물과 방법용으로 이용될 수 있다. AAV-TT 변이체 캡시드는 AAV2, AAV9 및 AAVrh10과 비교하여 CNS의 전역에서 증강된 향신경성 및 견실한 분포를 나타낸다. 유사하게, Hammond et al. (PLoS ONE 12(2):e0188830, 2017; 본원에서 온전히 참조로서 편입됨)에서 개시된 AAV-DJ8 벡터는 우수한 향신경성을 나타내고 본원 발명의 조성물과 방법용으로 적합할 수 있다.
특정 실례에서, 본원 발명은 (i) 조절 요소의 제어하에 ASO 서열 (예를 들면, 서열 번호: 1-100 중에서 하나)을 인코딩하는 폴리뉴클레오티드를 내포하고 ITR과 측접된 발현 카세트; 및 (ii) AAV9 캡시드 단백질의 아미노산 서열을 갖거나 또는 AAV9 캡시드의 생물학적 기능을 유지하면서 AAV9 캡시드 단백질의 아미노산 서열에 적어도 95%, 96%, 97%, 98%, 99% 또는 99.9% 동일한 바이러스 캡시드를 포함하는 인공 유전체를 포함하는 AAV9 벡터를 특징으로 한다. 인코딩된 AAV9 캡시드는 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 또는 30개의 아미노산 치환을 갖고 AAV9 캡시드의 생물학적 기능을 유지하는, 본원에서 온전히 참조로서 편입되는 U.S. 특허 번호 7,906,111에서 진술된 서열 번호: 116의 서열을 가질 수 있다.
(i) 조절 요소의 제어하에 폴리뉴클레오티드를 내포하고 ITR과 측접된 발현 카세트; 및 (ii) AAVrh10 캡시드 단백질의 아미노산 서열을 갖거나 또는 AAVrh10 캡시드의 생물학적 기능을 유지하면서 AAVrh10 캡시드 단백질의 아미노산 서열에 적어도 95%, 96%, 97%, 98%, 99% 또는 99.9% 동일한 바이러스 캡시드를 포함하는 인공 유전체를 포함하는 AAVrh10 벡터 역시 본원에서 제공된다. 인코딩된 AAVrh10 캡시드는 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 또는 30개의 아미노산 치환을 갖고 AAVrh10 캡시드의 생물학적 기능을 유지하는, 본원에서 온전히 참조로서 편입되는 U.S. 특허 번호 9,790,427에서 진술된 서열 번호: 81의 서열을 가질 수 있다.
유전자 조절 요소는 포유류 세포 (예를 들면, 뉴런)에서 기능적이도록 선택될 수 있다. 작동가능하게 연결된 구성요소를 내포하는 결과적인 작제물은 (5' 및 3') 기능적 AAV ITR 서열과 측접된다. 특정 실례는 포유류 CNS의 세포, 특히 뉴런에 대한 향성 및 이들에서 높은 형질도입 효율을 갖는 AAV 혈청형으로부터 유래된 벡터를 포함한다. 상이한 혈청형의 형질도입 효율의 검토 및 비교가 본 특허 출원에서 제공된다. 일정한 실례에서, AAV2, AAV5, AAV9 및 AAVrh10 기반 벡터는 예를 들면, 뉴런 및/또는 신경아교 세포를 형질도입함으로써 CNS에서 폴리뉴클레오티드의 장기간 발현을 주도한다.
AAV ITR과 측접된, 관심되는 폴리뉴클레오티드 (예를 들면, 본원에서 설명된 ASO 작용제를 인코딩하는 폴리뉴클레오티드)를 품는 AAV 발현 벡터는 선택된 서열(들)을, 주요 AAV 개방 해독틀 ("ORF")이 그로부터 절제된 AAV 유전체 내로 직접적으로 삽입함으로써 작제될 수 있다. 복제와 포장 기능을 가능하게 할 만큼 충분한 ITR의 부분이 남아있기만 하면, AAV 유전체의 다른 부분 역시 결실될 수 있다. 이런 작제물은 당해 분야에서 널리 알려진 기술을 이용하여 설계될 수 있다. 참조: 예를 들면, U.S. 특허 번호 5,173, 414 및 5,139, 941; 국제 공개 번호 WO 92/01070 (1992년 1월 23일 자 공개됨) 및 WO 93/03769 (1993년 3월 4일 자 공개됨). 대안으로, AAV ITR은 바이러스 유전체로부터 또는 이것을 내포하는 AAV 벡터로부터 절제될 수 있고, 그리고 표준 핵산 결찰 기술을 이용하여 다른 벡터 내에 존재하는 선택된 핵산 작제물의 5'와 3' 단부에 융합될 수 있다. ITR을 내포하는 AAV 벡터는 예를 들면, U.S. 특허 번호 5,139,941에서 설명되었다. 특히, 여러 AAV 벡터가 거기에서 설명되는데, 이들은 수탁 번호 53222, 53223, 53224, 53225 및 53226 하에 American Type Culture Collection ("ATCC")으로부터 가용하다. 추가적으로, 하나 이상의 선택된 핵산 서열에 상대적으로 5' 및 3'에 배열된 AAV ITR 서열을 포함하도록 키메라 유전자가 합성적으로 생산될 수 있다. 포유류 CNS 세포에서 키메라 유전자 서열의 발현을 위한 선호되는 코돈이 이용될 수 있고, 그리고 일정한 경우에, 폴리뉴클레오티드의 코돈 최적화가 널리 알려진 방법에 의해 수행될 수 있다. 완전한 키메라 서열이, 표준 방법에 의해 제조된 중첩되는 폴리뉴클레오티드로부터 조립된다. AAV 비리온을 생산하기 위해, 공지된 기술을 이용하여, 예컨대 형질감염에 의해 AAV 발현 벡터가 적합한 숙주 세포 내로 도입된다. 다양한 형질감염 기술이 당해 분야에서 전반적으로 알려져 있다. 특히 적합한 형질감염 방법은 인산칼슘 공동침전, 배양된 세포 내로 직접적인 미세주입, 전기천공, 리포솜 매개 유전자 전달, 지질 매개 형질도입, 그리고 고속 미세발사체를 이용한 핵산 전달을 포함한다.
예를 들면, 본원 발명의 특정 바이러스 벡터는 본원 발명의 핵산 서열 (예를 들면, 서열 번호: 1-100 중에서 하나)에 더하여, WPRE의 야생형 또는 돌연변이체 형태 및 토끼 베타-글로빈 폴리A 서열과 함께 또는 이들 없이, AAV2 바이러스로부터 유래된 ITR을 갖는 AAV 벡터 플라스미드의 백본, 프로모터 예컨대 예를 들면, U6 작은 핵 1 프로모터 또는 이의 변이체, H1 프로모터, 7SK 프로모터, ApoE-hAAT 프로모터, CBA 프로모터, CK8 프로모터, mU1a 프로모터, EF-1α 프로모터, TBG 프로모터, 뮤린 PGK 프로모터 또는 CAG 프로모터, 또는 임의의 뉴런 프로모터 예컨대 hSyn 프로모터, NeuN 프로모터, CaMKII 프로모터, Tα-1 프로모터, NSE 프로모터, PDGFβ 프로모터, VGLUT 프로모터, SST 프로모터, NPY 프로모터, VIP 프로모터, PV 프로모터, GAD65 또는 GAD67 프로모터, DRD1 프로모터, DRD2 프로모터, MAP1B 프로모터, C1ql2 프로모터, POMC 프로모터, 또는 Prox1 프로모터를 포함할 수 있다 (표 5 및 표 6 참조).
본원 발명은 (i) 조절 요소의 제어하에 폴리뉴클레오티드를 내포하고 ITR과 측접된 발현 카세트 및 (ii) AAV 캡시드를 포함하는 rAAV에 더욱 관계하는데, 여기서 상기 폴리뉴클레오티드는 Grik2 mRNA의 적어도 일부 또는 영역 (예를 들면, 서열 번호: 115-681에서 설명된 Grik2 mRNA의 일부 또는 영역 중에서 하나)에 특이적으로 결합하고 세포 (예를 들면, 뉴런)에서 GluK2 단백질의 발현을 저해하는 (예를 들면, 녹다운시키는) 저해성 RNA (예를 들면, ASO, 예컨대 예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA, 또는 shmiRNA, 그리고 특히, 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 ASO를 인코딩한다.
AAV 벡터는 예를 들면, Grik2 mRNA에 결합하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA, 또는 shmiRNA) 서열 및 hSyn 프로모터를 포함할 수 있다. 예를 들면, AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 서열 번호: 790 중에서 하나의 핵산 서열 또는 서열 번호: 682-685 또는 서열 번호: 790 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 hSyn 프로모터)를 내포할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 NeuN 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 NeuN 프로모터 (예를 들면, 서열 번호: 686의 핵산 서열 또는 서열 번호: 686의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 NeuN 프로모터)를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 CaMKII 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 서열 번호: 802 중에서 하나의 핵산 서열 또는 서열 번호: 687-691 및 서열 번호: 802 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 CaMKII 프로모터)를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 NSE 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 NSE 프로모터 (예를 들면, 서열 번호: 692 또는 693의 핵산 서열 또는 서열 번호: 서열 번호: 692 또는 693의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 NSE 프로모터)를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 PDGFβ 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 PDGFβ 프로모터 (예를 들면, 서열 번호: 694-696 중에서 하나의 핵산 서열 또는 서열 번호: 694-696 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 PDGFβ 프로모터)를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 VGluT 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 VGluT 프로모터 (예를 들면, 서열 번호: 697-701 중에서 하나의 핵산 서열 또는 서열 번호: 708-712 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 VGluT 프로모터)를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 SST 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 SST 프로모터 (예를 들면, 서열 번호: 702 또는 서열 번호: 703의 핵산 서열 또는 서열 번호: 702 또는 서열 번호: 703의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 SST 프로모터)를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 NPY 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 NPY 프로모터 (예를 들면, 서열 번호: 704의 핵산 서열 또는 서열 번호: 704의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 NPY 프로모터)를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 VIP 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 VIP 프로모터 (예를 들면, 서열 번호: 705 또는 서열 번호: 706의 핵산 서열 또는 서열 번호: 705 또는 서열 번호: 706의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 VIP 프로모터)를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 PV 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 PV 프로모터 (예를 들면, 서열 번호: 707-709 중에서 하나의 핵산 서열 또는 서열 번호: 707-709 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 PV 프로모터)를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 GAD65 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 GAD65 프로모터 (예를 들면, 서열 번호: 710-713 중에서 하나의 핵산 서열 또는 서열 번호: 710-713 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 GAD65 프로모터)를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 GAD67 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 GAD67 프로모터 (예를 들면, 서열 번호: 714 또는 서열 번호: 715의 핵산 서열 또는 서열 번호: 714 또는 서열 번호: 715의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 GAD67 프로모터)를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 DRD1 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 DRD1 프로모터 (예를 들면, 서열 번호: 716의 핵산 서열 또는 서열 번호: 716의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 DRD1 프로모터)를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 DRD2 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 DRD2 프로모터 (예를 들면, 서열 번호: 717 또는 서열 번호: 718의 핵산 서열 또는 서열 번호: 717 또는 서열 번호: 718의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 DRD2 프로모터)를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 C1ql2 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791의 핵산 서열 또는 서열 번호: 719 또는 서열 번호: 791의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 C1ql2 프로모터)를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 POMC 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 POMC 프로모터 (예를 들면, 서열 번호: 720의 핵산 서열 또는 서열 번호: 720의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 POMC 프로모터)를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 PROX1 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 PROX1 프로모터 (예를 들면, 서열 번호: 721 또는 서열 번호: 722의 핵산 서열 또는 서열 번호: 721 또는 서열 번호: 722의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 PROX1 프로모터)를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 MAP1B 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 MAP1B 프로모터 (예를 들면, 서열 번호: 723-725 중에서 하나의 핵산 서열 또는 서열 번호: 723-725 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 MAP1B 프로모터)를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 Tα-1 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 Tα-1 프로모터 (예를 들면, 서열 번호: 726 또는 서열 번호: 727의 핵산 서열 또는 서열 번호: 726 또는 서열 번호: 727의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 Tα-1 프로모터)를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 U6 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 U6 프로모터, 예컨대, 서열 번호: 728-733 또는 772 중에서 하나의 핵산 서열 또는 서열 번호: 728-733 또는 772 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 U6 프로모터를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 H1 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 H1 프로모터, 예컨대, 서열 번호: 734의 핵산 서열 또는 서열 번호: 734의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 H1 프로모터를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 7SK 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 7SK 프로모터, 예컨대, 서열 번호: 735의 핵산 서열 또는 서열 번호: 735의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 7SK 프로모터를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 ApoE-hAAT 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 ApoE-hAAT 프로모터, 예컨대, 서열 번호: 736의 핵산 서열 또는 서열 번호: 736의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 ApoE-hAAT 프로모터를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 CAG 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 CAG 프로모터, 예컨대, 서열 번호: 737의 핵산 서열 또는 서열 번호: 737의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 CAG 프로모터를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 CBA 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 CBA 프로모터, 예컨대, 서열 번호: 738의 핵산 서열 또는 서열 번호: 738의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 CBA 프로모터를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 CK8 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 CK8 프로모터, 예컨대, 서열 번호: 739의 핵산 서열 또는 서열 번호: 739의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 CK8 프로모터를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 mU1a 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 mU1a 프로모터, 예컨대, 서열 번호: 740의 핵산 서열 또는 서열 번호: 740의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 mU1a 프로모터를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 EF-1α 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 EF-1α 프로모터, 예컨대, 서열 번호: 741의 핵산 서열 또는 서열 번호: 741의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 EF-1α 프로모터를 포함할 수 있다.
대안으로, AAV 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 TBG 프로모터를 포함할 수 있다. 예를 들면, 개시된 AAV 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 TBG 프로모터, 예컨대, 서열 번호: 742의 핵산 서열 또는 서열 번호: 742의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 TBG 프로모터를 포함할 수 있다.
레트로바이러스 벡터
본원에서 설명된 방법과 조성물에서 이용된 전달 벡터는 레트로바이러스 벡터일 수 있다. 본원에서 설명된 방법과 조성물에서 이용될 수 있는 레트로바이러스 벡터의 한 가지 유형은 렌티바이러스 벡터이다. 레트로바이러스의 부분집합인 렌티바이러스 벡터 (LV)는 넓은 범위의 분열과 비분열 세포 유형을 높은 효율로 형질도입하여, 폴리뉴클레오티드의 안정된, 장기간 발현을 부여한다. LV를 포장하고 형질도입하기 위한 최적화 전략의 개요는 Delenda, The Journal of Gene Medicine 6: S125 (2004)에서 제공되는데, 이것의 개시는 본원에서 참조로서 편입된다.
렌티바이러스 기반 유전자 전달 기술의 이용은 관심되는 폴리뉴클레오티드가 수용되는 고도로 결실된 바이러스 유전체를 보유하는 재조합 렌티바이러스 입자의 시험관내 생산에 의존한다. 특히, 재조합 렌티바이러스는 (1) 포장 작제물, 다시 말하면, Rev와 함께 Gag-Pol 전구체를 발현하는 (대안으로 트랜스로 발현된) 벡터; (2) 일반적으로 이종성 성질의 외피 수용체를 발현하는 벡터; 그리고 (3) 모든 개방 해독틀이 박탈되지만, 복제, 캡시드화 및 발현을 위해 필요한 서열을 유지하는 바이러스 cDNA에 있고, 발현되는 서열이 삽입되는 전달 벡터의 허용적 세포주에서 트랜스 공동발현을 통해 회수된다.
본원에서 설명된 방법과 조성물에서 이용된 LV는 5'-긴 말단 반복 (LTR), HIV 신호 서열, HIV Psi 신호 5'-스플라이스 부위 (SD), 델타-GAG 요소, Rev 반응 요소 (RRE), 3'-스플라이스 부위 (SA), 신장 인자 (EF) 1-알파 프로모터 및 3'-자가 비활성화 LTR (SIN-LTR) 중에서 하나 이상을 포함할 수 있다. 렌티바이러스 벡터는 임의적으로, US 6,136,597에서 설명된 바와 같은 중심 폴리퓨린 관 (cPPT) 및 우드척 간염 바이러스 전사후 조절 요소 (WPRE)를 포함하고, 이것의 개시는 이것이 WPRE와 관련되는 한, 본원에서 참조로서 편입된다. 렌티바이러스 벡터는 pHR' 백본을 더욱 포함할 수 있는데, 이것은 예를 들면 아래에 제시된 바와 같이 포함할 수 있다.
Lu et al., Journal of Gene Medicine 6:963 (2004)에서 설명된 Lentigen LV가 DNA 분자를 발현하고 및/또는 세포를 형질도입하는 데 이용될 수 있다. 본원에서 설명된 방법과 조성물에서 이용된 LV는 5'-긴 말단 반복 (LTR), HIV 신호 서열, HIV Psi 신호 5'-스플라이스 부위 (SD), 델타-GAG 요소, Rev 반응 요소 (RRE), 3'-스플라이스 부위 (SA), 신장 인자 (EF) 1-알파 프로모터 및 3'-자가 비활성화 LTR (SIN-LTR)을 포함할 수 있다. 임의적으로, 이들 영역 중에서 하나 이상이 유사한 기능을 수행하는 다른 영역으로 치환된다.
인핸서 요소가 변형된 DNA 분자의 발현을 증가시키거나 또는 렌티바이러스 통합 효율을 증가시키는 데 이용될 수 있다. 본원에서 설명된 방법과 조성물에서 이용된 LV는 nef 서열을 포함할 수 있다. 본원에서 설명된 방법과 조성물에서 이용된 LV는 벡터 통합을 증강하는 cPPT 서열을 포함할 수 있다. cPPT는 (+)-가닥 DNA 합성의 두 번째 기원으로서 행동하고 자신의 선천적 HIV 유전체의 중간에 부분적인 가닥 중첩을 도입한다. 전달 벡터 백본에서 cPPT 서열의 도입은 핵 수송 및 표적 세포의 DNA 내로 통합된 유전체의 총량을 강하게 증가시켰다. 본원에서 설명된 방법과 조성물에서 이용된 LV는 우드척 전사후 조절 요소 (WPRE)를 포함할 수 있다. WPRE는 전사체의 핵 이출을 증진함으로써 및/또는 미성숙 전사체의 폴리아데닐화의 효율을 증가시켜, 세포에서 mRNA의 총량을 증가시킴으로써, 전사 수준에서 기능한다. LV에 WPRE의 부가는 시험관내와 생체내 둘 모두에서 여러 상이한 프로모터로부터 폴리뉴클레오티드 발현의 수준에서 실제적인 향상을 야기한다. 본원에서 설명된 방법과 조성물에서 이용된 LV는 cPPT 서열 및 WPRE 서열 둘 모두를 포함할 수 있다. 벡터는 또한, 단일 프로모터로부터 다중 폴리펩티드의 발현을 허용하는 IRES 서열을 포함할 수 있다.
IRES 서열 이외에, 복수의 폴리뉴클레오티드의 발현을 허용하는 다른 요소가 유용하다. 본원에서 설명된 방법과 조성물에서 이용된 벡터는 하나 초과의 폴리뉴클레오티드의 발현을 허용하는 복수의 프로모터를 포함할 수 있다. 장래에 확인될 복수의 폴리뉴클레오티드의 발현을 허용하는 다른 요소가 유용하고, 그리고 본원에서 설명된 조성물과 방법용으로 적합한 벡터에서 활용될 수 있다. 본원에서 설명된 방법과 조성물에서 이용된 벡터는 임상 등급 벡터일 수 있다.
따라서, 레트로바이러스 벡터가 개시된 방법과 조성물용으로 이용될 수 있다. 레트로바이러스는 그들의 유전자를 숙주 유전체 내로 통합하고, 대량의 외래 유전 물질을 전달하고, 광역의 종 및 세포 유형을 감염시키고, 그리고 특수한 세포주에 포장되는 능력으로 인해 유전자 전달 벡터로서 선택될 수 있다. 레트로바이러스 벡터를 작제하기 위해, 복제 결함성인 바이러스를 생산하기 위해 특정한 바이러스 서열 대신에 관심되는 유전자를 인코딩하는 핵산이 바이러스 유전체 내로 삽입된다. 비리온을 생산하기 위해, gag, pol 및/또는 env 유전자를 내포하지만 LTR 및 포장 구성요소가 없는 포장 세포주가 작제된다. 레트로바이러스 LTR 및 포장 서열과 함께, cDNA를 내포하는 재조합 플라스미드가 이러한 세포주 내로 도입될 때 (예를 들면, 인산칼슘 침전에 의해), 포장 서열은 재조합 플라스미드의 RNA 전사체가 바이러스 입자 내로 포장되도록 허용하고, 이들 바이러스 입자는 이후, 배양 배지 내로 분비된다. 이후, 재조합 레트로바이러스를 내포하는 배지가 수집되고, 임의적으로 농축되고, 그리고 유전자 전달에 이용된다. 레트로바이러스 벡터는 광범위한 다양한 세포 유형을 감염시킬 수 있다.
추가적으로, 렌티바이러스 벡터가 본원에서 개시된 방법과 조성물용으로 이용될 수 있다. 따라서, 본원 발명의 목적은 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA, 또는 shmiRNA) 서열 (예를 들면, 서열 번호: 1-100에서 설명된 ASO 서열 중에서 하나)을 포함하는 렌티바이러스 벡터에 관계한다.
따라서, 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 포함할 수 있다. 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO 서열 (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA, 또는 shmiRNA) 및 hsyn 프로모터를 포함할 수 있다.
렌티바이러스 벡터는 예를 들면, Grik2 mRNA에 결합하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA, 또는 shmiRNA) 서열 및 hSyn 프로모터를 포함할 수 있다. 예를 들면, 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 서열 번호: 790 중에서 하나의 핵산 서열 또는 서열 번호: 682-685 및 서열 번호: 790 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 hSyn 프로모터)를 내포할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 NeuN 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 NeuN 프로모터 (예를 들면, 서열 번호: 686의 핵산 서열 또는 서열 번호: 686의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 NeuN 프로모터)를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 CaMKII 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 서열 번호: 802 중에서 하나의 핵산 서열 또는 서열 번호: 687-691 및 서열 번호: 802 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 CaMKII 프로모터)를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 NSE 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 NSE 프로모터 (예를 들면, 서열 번호: 692 또는 서열 번호: 693의 핵산 서열 또는 서열 번호: 서열 번호: 692 또는 서열 번호: 693의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 NSE 프로모터)를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 PDGFβ 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 PDGFβ 프로모터 (예를 들면, 서열 번호: 694-696 중에서 하나의 핵산 서열 또는 서열 번호: 694-696 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 PDGFβ 프로모터)를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 VGluT 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 VGluT 프로모터 (예를 들면, 서열 번호: 697-701 중에서 하나의 핵산 서열 또는 서열 번호: 697-701 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 VGluT 프로모터)를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 SST 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 SST 프로모터 (예를 들면, 서열 번호: 702 또는 서열 번호: 703 중에서 하나의 핵산 서열 또는 서열 번호: 702 또는 서열 번호: 703 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 SST 프로모터)를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 NPY 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 NPY 프로모터 (예를 들면, 서열 번호: 704의 핵산 서열 또는 서열 번호: 704 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 NPY 프로모터)를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 VIP 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 VIP 프로모터 (예를 들면, 서열 번호: 705 또는 서열 번호: 706의 핵산 서열 또는 서열 번호: 705 또는 서열 번호: 706 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 VIP 프로모터)를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 PV 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 PV 프로모터 (예를 들면, 서열 번호: 707-709 중에서 하나의 핵산 서열 또는 서열 번호: 707-709 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 PV 프로모터)를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 GAD65 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 GAD65 프로모터 (예를 들면, 서열 번호: 710-713 중에서 하나의 핵산 서열 또는 서열 번호: 710-713 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 GAD65 프로모터)를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 GAD67 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 GAD67 프로모터 (예를 들면, 서열 번호: 714 또는 서열 번호: 715의 핵산 서열 또는 서열 번호: 714 또는 서열 번호: 715 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 GAD67 프로모터)를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 DRD1 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 DRD1 프로모터 (예를 들면, 서열 번호: 716의 핵산 서열 또는 서열 번호: 716 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 DRD1 프로모터)를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 DRD2 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 DRD2 프로모터 (예를 들면, 서열 번호: 717 또는 서열 번호: 718의 핵산 서열 또는 서열 번호: 717 또는 서열 번호: 718 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 DRD2 프로모터)를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 C1ql2 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791의 핵산 서열 또는 서열 번호: 719 또는 서열 번호: 791의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 C1ql2 프로모터)를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 POMC 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 POMC 프로모터 (예를 들면, 서열 번호: 720의 핵산 서열 또는 서열 번호: 720 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 POMC 프로모터)를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 PROX1 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 PROX1 프로모터 (예를 들면, 서열 번호: 721 또는 서열 번호: 722의 핵산 서열 또는 서열 번호: 721 또는 서열 번호: 722 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 PROX1 프로모터)를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 MAP1B 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 MAP1B 프로모터 (예를 들면, 서열 번호: 723-725 중에서 하나의 핵산 서열 또는 서열 번호: 723-725 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 MAP1B 프로모터)를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 Tα-1 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 Tα-1 프로모터 (예를 들면, 서열 번호: 726 또는 서열 번호: 727의 핵산 서열 또는 서열 번호: 726 또는 서열 번호: 727 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 Tα-1 프로모터)를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 U6 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 U6 프로모터, 예컨대, 서열 번호: 728-733 또는 772 중에서 하나의 핵산 서열 또는 서열 번호: 728-733 또는 772 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 U6 프로모터를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 H1 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 H1 프로모터, 예컨대, 서열 번호: 734 중에서 하나의 핵산 서열 또는 서열 번호: 734의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 H1 프로모터를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 7SK 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 7SK 프로모터, 예컨대, 서열 번호: 735의 핵산 서열 또는 서열 번호: 735의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 7SK 프로모터를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 ApoE-hAAT 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 ApoE-hAAT 프로모터, 예컨대, 서열 번호: 736의 핵산 서열 또는 서열 번호: 736의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 ApoE-hAAT 프로모터를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 CAG 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 CAG 프로모터, 예컨대, 서열 번호: 737의 핵산 서열 또는 서열 번호: 737의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 CAG 프로모터를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 CBA 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 CBA 프로모터, 예컨대, 서열 번호: 738의 핵산 서열 또는 서열 번호: 738의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 CBA 프로모터를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 CK8 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 CK8 프로모터, 예컨대, 서열 번호: 739의 핵산 서열 또는 서열 번호: 739의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 CK8 프로모터를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 mU1a 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 mU1a 프로모터, 예컨대, 서열 번호: 740의 핵산 서열 또는 서열 번호: 740의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 mU1a 프로모터를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 EF-1α 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 EF-1α 프로모터, 예컨대, 서열 번호: 741의 핵산 서열 또는 서열 번호: 741의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 EF-1α 프로모터를 포함할 수 있다.
대안으로, 렌티바이러스 벡터는 Grik2 mRNA에 결합하고 이것의 발현을 저해하는 ASO (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA) 서열 및 TBG 프로모터를 포함할 수 있다. 예를 들면, 개시된 렌티바이러스 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체, 그리고 TBG 프로모터, 예컨대, 서열 번호: 742의 핵산 서열 또는 서열 번호: 742의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 TBG 프로모터를 포함할 수 있다.
렌티바이러스는 복합적인 레트로바이러스인데, 이들은 통상적인 레트로바이러스 유전자 gag, pol 및 env 이외에, 조절 또는 구조 기능을 갖는 다른 유전자를 내포한다. 더 높은 복잡성은 바이러스가 잠복 감염의 경과의 경우에서와 같이, 자신의 생활 주기를 조정하는 것을 가능하게 한다. 렌티바이러스의 일부 실례는 인간 면역결핍 바이러스 (HIV1, HIV2) 및 유인원 면역결핍 바이러스 (SIV)를 포함한다. 렌티바이러스 벡터는 HIV 독력 유전자를 여러 번 약화시킴으로써 생성되었다, 예를 들면, 유전자 env, vif, vpr, vpu 및 nef가 결실되어 벡터가 생물학적으로 안전해진다. 렌티바이러스 벡터는 당해 분야에서 공지된다, 참조: 예를 들면, U.S. 특허 번호 6,013,516 및 5,994,136, 이들 둘 모두 본원에서 참조로서 편입된다. 일반적으로, 벡터는 플라스미드 기반 또는 바이러스 기반이고, 그리고 외래 핵산의 통합 및 선택 및 상기 핵산의 숙주 세포 내로의 전달을 위한 필수 서열을 보유하도록 설정된다. 관심되는 벡터의 gag, pol 및 env 유전자 또한 당해 분야에서 공지된다. 따라서, 유관한 유전자는 선택된 벡터 내로 클로닝되고, 이후 관심되는 표적 세포를 형질전환하는 데 이용된다. 비분열 세포를 감염시킬 수 있는 재조합 렌티바이러스 (여기서 적합한 숙주 세포가 포장 단백질, 다시 말하면 gag, pol 및 env뿐만 아니라 rev 및 tat를 보유하는 두 개 이상의 벡터로 형질감염된다)는 본원에서 참조로서 편입되는 U.S. 특허 번호 5,994,136에서 설명된다. 이 공보는 바이러스 gag 및 pol 유전자를 인코딩하는 핵산을 제공할 수 있는 첫 번째 벡터, 그리고 포장 세포를 생산하기 위한 바이러스 env를 인코딩하는 핵산을 제공할 수 있는 두 번째 벡터를 제공한다. 이종성 유전자를 제공하는 벡터를 상기 포장 세포 내로 도입하는 것은 관심되는 외래 유전자를 보유하는 감염성 바이러스 입자를 방출하는 생산자 세포를 생성한다. env는 인간 및 다른 종의 세포의 형질도입을 가능하게 하는 암포트로픽 외피 단백질일 수 있다. 전형적으로, 본원 발명의 핵산 분자 또는 벡터는 "제어 서열"을 포함하는데, 이것은 수용자 세포에서 코딩 서열의 복제, 전사 및 번역을 집합적으로 제공하는 프로모터 서열, 폴리아데닐화 신호, 전사 종결 서열, 상류 조절 도메인, 복제 기점, 내부 리보솜 유입 부위 ("IRES"), 인핸서 등을 집합적으로 지칭한다. 선별된 코딩 서열이 적절한 숙주 세포에서 복제되고, 전사되고, 번역될 수 있기만 하면, 이들 제어 서열 모두가 항상 존재할 필요는 없다.
바이러스 조절 요소
바이러스 조절 요소는 핵산 분자를 숙주 세포 내로 도입하는 데 이용되는 전달 수송체의 구성요소이다. 바이러스 조절 요소는 임의적으로, 레트로바이러스 조절 요소이다. 예를 들면, 바이러스 조절 요소는 HSC1 또는 MSCV로부터 유래된 LTR 및 gag 서열일 수 있다. 레트로바이러스 조절 요소는 렌티바이러스로부터 유래될 수 있거나 또는 이들은 다른 유전체 영역으로부터 확인된 이종성 서열일 수 있다. 다른 바이러스 조절 요소가 알려짐에 따라서, 이들은 본원에서 설명된 방법과 조성물용으로 이용될 수 있다.
Grik2 안티센스 올리고뉴클레오티드를 인코딩하는 바이러스 벡터
본원 발명은 이종성 폴리뉴클레오티드의 전달을 위한 핵산 벡터에 관계하는데, 여기서 상기 폴리뉴클레오티드는 Grik2 mRNA에 특이적으로 결합하고 세포에서 GluK2 단백질의 발현을 저해하는 저해성 ASO 작용제 (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA, 또는 shmiRNA) 작제물을 인코딩한다. 따라서, 본원 발명의 목적은 적어도 Grik2 mRNA의 영역 또는 일부 (예를 들면, 서열 번호: 115-681 중에서 하나, 또는 서열 번호: 115-681 중에서 하나의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에서 선택되는 Grik2 mRNA의 영역 또는 일부 중에서 하나에 완전히 또는 실제적으로 상보적인 올리고뉴클레오티드 서열을 포함하는 벡터를 제공한다. 본원 발명의 벡터는 Grik2 mRNA의 하나 이상의 영역에 완전히 또는 실제적으로 상보적인 올리고뉴클레오티드 서열의 임의의 변이체를 포함할 수 있다. 추가적으로, 본원 발명의 벡터는 GluK2 단백질의 임의의 변이체를 인코딩하는 Grik2 mRNA에 완전히 또는 실제적으로 상보적인 올리고뉴클레오티드 서열의 임의의 변이체를 포함할 수 있다.
따라서, 관심되는 이중 가닥 RNA를 인코딩하는 DNA는 유전자 카세트, 예를 들면 DNA의 전사가 프로모터 및/또는 다른 조절 요소에 의해 제어되는 발현 카세트 내로 통합된다. DNA는 관심되는 Grik2 ASO (예를 들면, 서열 번호: 1-100 중에서 하나)를 발현하는 벡터의 이런 발현 카세트 내로 통합되고, 그리고 표적 세포에 전달을 위한 관심되는 바이러스 벡터에 의해 캡시드에 싸인다. 본원 발명의 바이러스 벡터는 따라서, 임의의 Grik2 mRNA 전사체 동종형 (예를 들면, 서열 번호: 115-124 중에서 하나)에 혼성화하는 임의의 안티센스 RNA를 인코딩한다. 바이러스 벡터는 예를 들면, 표 2 또는 표 3에서 열거된 siRNA 중에서 하나를 인코딩한다.
본원 발명의 벡터는 Grik2 mRNA의 적어도 일부 또는 영역 (예를 들면, 서열 번호: 115-681에서 설명된 Grik2 mRNA, 또는 서열 번호: 115-681 중에서 하나의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체의 영역 또는 일부 중에서 하나)을 인식하거나 또는 이것에 결합하는 ASO를 인코딩하는 폴리뉴클레오티드를 전달한다. ASO 작용제를 인코딩하는 이종성 폴리뉴클레오티드는 세포 (예를 들면, 포유류 세포, 예컨대 예를 들면, 인간 세포, 예컨대 예를 들면, 뉴런 세포, 예컨대 예를 들면, DGC) 내에 이런 ASO의 처리를 담보하는 더 큰 작제물 또는 골격의 일부일 수 있다. 표 2 또는 표 3에서 열거된 siRNA 중에서 하나를 인코딩하는 폴리뉴클레오티드는 마이크로RNA 유전자 (예를 들면, 그 중에서도 특히, miR-30, miR-155, miR-281-1, 또는 miR-124-3)의 전구체 또는 일부, 예컨대 예를 들면, 마이크로RNA 유전자의 5' 측부 서열, 3' 측부 서열, 또는 루프 서열을 포함할 수 있다.
따라서, 본원 발명의 목적은 이종성 폴리뉴클레오티드를 포함하고, 그리고 5 '에서 3'로, 예를 들면, 프로모터 (예를 들면, 표 5 및 표 6에서 설명된 프로모터 중에서 하나), 임의적으로 인트론 (예를 들면, 표 7에서 설명된 인트론 중에서 하나), Grik2 mRNA 발현을 저해하는 ASO 작용제 (예를 들면, 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 ASO 작용제)를 인코딩하는 뉴클레오티드 서열, 그리고 폴리A 서열 (예를 들면, 표 7에서 설명된 폴리A 서열 중에서 하나)을 내포하는 발현 벡터에 관계한다. 발현 벡터는 또한, 5' 반전 말단 반복 (ITR)에서 3' ITR로, 5' ITR (예를 들면, 표 7에서 설명된 5' 또는 3' ITR 서열 중에서 하나), 프로모터, 임의적으로 인트론, Grik2 mRNA 발현을 저해하는 ASO를 인코딩하는 뉴클레오티드 서열, 폴리A 서열, 그리고 3' ITR을 포함할 수 있다. 발현 벡터는 임의의 전술한 벡터 요소에 인접된 스페이서 및/또는 링커 서열을 더욱 내포할 수 있다.
특정 실례에서, 발현 벡터 또는 폴리뉴클레오티드는 스템 루프 구조를 형성하는 스템 및 루프를 인코딩하는 뉴클레오티드 서열을 포함할 수 있는데, 여기서 상기 루프는 표 2 또는 표 3에서 열거된 ASO 작용제 중에서 하나를 인코딩하는 뉴클레오티드 서열을 포함한다. 예를 들면, 발현 벡터 또는 폴리뉴클레오티드는 루프 영역을 인코딩하는 핵산 서열을 포함할 수 있는데, 여기서 상기 루프 영역은 야생형 마이크로RNA 서열 유전자 (예를 들면, 그 중에서도 특히, miR-30, miR-155, miR-281-1, 또는 miR-124-3)로부터 전체적으로 또는 부분적으로 유래될 수 있거나 또는 완전히 인공일 수 있다. 특정 실례에서, 루프 영역은 miR-30a 루프 서열일 수 있다. 게다가, 스템 루프 구조는 가이드 서열 (예를 들면, 안티센스 RNA 서열, 예컨대 예를 들면, 서열 번호: 1-100 중에서 하나) 및 가이드 서열의 전부 또는 일부에 상보적인 승객 서열을 포함할 수 있다. 예를 들면, 승객 서열은 가이드 서열의 10, 9, 8, 7, 6, 5, 4, 3, 2 또는 1개의 뉴클레오티드(들)를 제외하고, 가이드 서열의 모든 뉴클레오티드에 상보적일 수 있거나, 또는 승객 서열은 서열 번호: 1-100 중에서 하나에 상보적일 수 있다.
프리-miRNA 또는 일차-miRNA 골격은 본원 발명의 가이드 (다시 말하면, 안티센스) 서열을 포함한다. 일차-miRNA 골격은 프리-miRNA 골격을 포함하고, 그리고 일차-miRNA는 길이에서 50-800개의 뉴클레오티드 (예를 들면, 50-800, 75-700, 100-600, 150-500, 200-400, 또는 250-300개의 뉴클레오티드)일 수 있다. 특정 실례에서, 프리-mRNA는 50-100개의 뉴클레오티드 (예를 들면, 50-60, 60-70, 70-80, 80-90, 또는 90-100개 사이의 뉴클레오티드), 100-200개의 뉴클레오티드 (예를 들면, 110-120, 120-130, 130-140, 140-150, 150-160, 160-170, 170-180, 180-190, 또는 190-200개 사이의 뉴클레오티드), 200-300개의 뉴클레오티드 (예를 들면, 200-210, 210-220, 220-230, 230-240, 240-250, 250-260, 260-270, 270-280, 280-290, 또는 290-300개 사이의 뉴클레오티드), 300-400개의 뉴클레오티드 (예를 들면, 300-310, 310-320, 320-330, 330-340, 340-350, 350-360, 360-370, 370-380, 380-390, 또는 390-400개 사이의 뉴클레오티드), 400-500개의 뉴클레오티드 (예를 들면, 400-410, 410-420, 420-430, 430-440, 440-450, 450-460, 460-470, 470-480, 480-490, 또는 490-500개 사이의 뉴클레오티드), 500-600개의 뉴클레오티드 (예를 들면, 500-510, 510-520, 520-530, 530-540, 540-550, 550-560, 560-570, 570-580, 580-590, 또는 590-600개 사이의 뉴클레오티드), 600-700개의 뉴클레오티드 (예를 들면, 600-610, 610-620, 620-630, 630-640, 640-650, 650-660, 660-670, 670-680, 680-690, 또는 690-700개 사이의 뉴클레오티드), 또는 700-800개의 뉴클레오티드 (예를 들면, 700-710, 710-720, 720-730, 730-740, 740-750, 750-760, 760-770, 770-780, 780-790, 또는 790-800개 사이의 뉴클레오티드)일 수 있다. 이들 조작된 골격은 프리-miRNA를, 가이드 가닥 및 승객 가닥을 포함하는 이중 가닥 RNA으로 처리하는 것을 가능하게 한다. 따라서, 프리-miRNA는 가이드 (다시 말하면, 안티센스) RNA를 인코딩하는 서열을 포함하는 5' 팔, 통상적으로 야생형 miRNA (예를 들면, 그 중에서도 특히, miR-30, miR-155, miR-281-1, 또는 miR-124-3)로부터 유래되는 루프 서열, 그리고 가이드 가닥에 완전히 또는 실제적으로 상보적인 승객 (다시 말하면, 센스) 가닥을 인코딩하는 서열을 포함하는 3' 팔을 포함한다. 프리-miRNA "스템 루프" 구조는 일반적으로, 길이에서 50개보다 긴 뉴클레오티드, 예를 들면 50-150개의 뉴클레오티드 (예를 들면, 50-60, 60-70, 70-80, 80-90, 90-100, 100-110, 110-120, 120-130, 130-140, 또는 140-150개의 뉴클레오티드), 50-110개의 뉴클레오티드 (예를 들면, 50-60, 60-70, 70-80, 80-90, 90-100, 100-110개의 뉴클레오티드), 또는 50-80개의 뉴클레오티드 (예를 들면, 50-60, 60-70, 70-80개의 뉴클레오티드)이다. 일차-miRNA는 5'와 3' 팔에 각각 측접하는 5' 측부 및 3' 측부 서열을 더욱 포함한다. 측접 서열은 다른 서열 (팔 영역 또는 가이드 서열)과 반드시 인접하는 것은 아니고, 비구조화, 비대합 영역이고, 그리고 또한, 하나 이상의 야생형 일차-miRNA 골격 (예를 들면, 그 중에서도 특히, miR-30, miR-155, miR-281-1, 또는 miR-124-3으로부터 전체적으로 또는 부분적으로 유래된 일차-miRNA 골격)으로부터 전체적으로 또는 부분적으로 유래될 수 있다. 측접 서열은 각각, 길이에서 적어도 4개의 뉴클레오티드, 또는 길이에서 300개까지의 뉴클레오티드 또는 그 초과 (예를 들면, 4-300, 10-275, 20-250, 30-225, 40-200, 50-175, 60-150, 70-125, 80-100, 또는 90-95개의 뉴클레오티드)이다. 스페이서 서열은 전술된 서열 구조 사이에 개재성으로 존재할 수 있고, 그리고 많은 경우에 연결 폴리뉴클레오티드, 예를 들면, 1-30개의 뉴클레오티드 (예를 들면, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 또는 30개의 뉴클레오티드)를 제공하여, 전체 프리-miRNA 구조에 대한 기능성을 간섭하지 않으면서 유연성을 제공한다. 스페이서는 스페이서가 이중 가닥 RNA의 처리를 간섭하지 않고 스페이서가 가이드 RNA의 표적 mRNA 서열과의 결합/상호작용을 간섭하지 않기만 하면, 자연 발생 연결기, 자연 발생 RNA, 자연 발생 연결기의 일부, 폴리-A 또는 폴리-U, 또는 뉴클레오티드의 무작위 서열로부터 유래될 수 있다.
본원에서 개시된 방법과 조성물에 따라서, 뉴클레오티드 서열을 포함하는 발현 벡터 또는 폴리뉴클레오티드는 (i) 가이드 (예를 들면, 안티센스) 가닥 및 임의적으로, 5' 스페이서 서열을 포함하는 5' 스템 루프 팔; 그리고 (ii) 승객 (예를 들면, 센스) 가닥 및 임의적으로 3' 스페이서 서열을 포함하는 3' 스템 루프 팔을 더욱 인코딩할 수 있다. 다른 실례에서, 뉴클레오티드 서열을 포함하는 발현 벡터 또는 폴리뉴클레오티드는 (i) 승객 가닥 및 임의적으로, 5' 스페이서 서열을 포함하는 5' 스템 루프 팔; 그리고 (ii) 가이드 가닥 및 임의적으로 3' 스페이서 서열을 포함하는 3' 스템 루프 팔을 더욱 인코딩할 수 있다. 다른 실례에서, 우리딘 동요 염기가 가이드 가닥의 5' 단부에 존재한다. 추가 실례에서, 발현 벡터 또는 폴리뉴클레오티드는 가이드 서열의 상류에 선도적인 5' 측부 영역을 포함하고, 그리고 상기 측부 영역은 임의의 길이일 수 있고, 그리고 야생형 마이크로RNA 서열로부터 전체적으로 또는 부분적으로 유래될 수 있거나, 이종성이거나 또는 다른 측부 영역 또는 루프로부터 상이한 기원의 miRNA로부터 유래될 수 있거나, 또는 완전히 인공일 수 있다. 3' 측부 영역은 크기 및 기원에서 5' 측부 영역을 반영할 수 있고, 그리고 3' 측부 영역은 가이드 서열의 하류 (다시 말하면, 3')일 수 있다. 또 다른 실례에서, 5' 측부 서열 및 3' 측부 서열 중에서 하나 또는 둘 모두 부재한다.
발현 벡터 또는 폴리뉴클레오티드는 첫 번째 측부 영역 (예를 들면, 표 8에서 설명된 5' 측부 영역 중에서 하나)을 더욱 인코딩하는 뉴클레오티드 서열을 포함할 수 있는데, 상기 첫 번째 측부 영역은 5' 측부 서열 및 임의적으로, 5' 스페이서 서열을 포함한다. 특정 실례에서, 첫 번째 측부 영역은 상기 승객 가닥의 상류 (다시 말하면, 5')에 위치된다. 다른 실례에서, 뉴클레오티드 서열을 포함하는 발현 벡터 또는 폴리뉴클레오티드는 두 번째 측부 영역 (예를 들면, 표 8에서 설명된 3' 측부 영역 중에서 하나)을 인코딩하는데, 상기 두 번째 측부 영역은 3' 측부 서열 및 임의적으로, 3' 스페이서 서열을 포함한다. 특정 실례에서, 첫 번째 측부 영역은 가이드 가닥의 5'에 위치된다.
본원에서 개시된 방법과 조성물에 따라서, 발현 벡터 또는 폴리뉴클레오티드는 다음을 인코딩하는 뉴클레오티드 서열을 포함할 수 있고:
(a) 스템 루프 서열로서, 5'에서 3'로:
(i) 표 2 또는 표 3에서 열거된 ASO 서열 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체) 중에서 하나에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과) 동일한 가이드 뉴클레오티드 서열을 포함하는 5' 스템 루프 팔;
(ii) 마이크로RNA 루프 영역으로서, 여기서 상기 루프 영역은 마이크로RNA 루프 서열 (예를 들면, miR-30a, miR-155, miR-218-1, 또는 miR-124-3 루프 서열 (예를 들면, 서열 번호: 758, 764, 767 또는 770 중 하나에서 선택된 핵산을 갖는 마이크로RNA 루프 서열)을 포함하고;
(iii) 가이드 가닥에 상보적이거나 또는 실질적으로 상보적인 승객 뉴클레오티드 서열을 포함하는 3' 스템 루프 팔을 포함하는 스템 루프 서열,
(b) 가이드 가닥의 5'에 위치된 첫 번째 측부 영역 (예를 들면, 표 8에서 설명된 5' 측부 영역 중에서 하나)으로서, 여기서 상기 첫 번째 측부 영역은 5' 측부 서열 및 임의적으로, 5' 스페이서 서열을 포함하고; 그리고
(c) 승객 가닥의 3'에 위치된 두 번째 측부 영역 (예를 들면, 표 8에서 설명된 3' 측부 영역 중에서 하나)으로서, 여기서 상기 두 번째 측부 영역은 3' 측부 서열 및 임의적으로, 3' 스페이서 서열을 포함한다.
다른 실례에서, 발현 벡터 또는 폴리뉴클레오티드는 다음을 인코딩하는 뉴클레오티드 서열을 포함하고:
(a) 스템 루프 서열로서, 5'에서 3'로:
(i) 가이드 뉴클레오티드 서열에 상보적이거나 또는 실질적으로 상보적인 승객 뉴클레오티드 서열을 포함하는 5' 스템 루프 팔;
(ii) 마이크로RNA 루프 영역으로서, 여기서 상기 루프 영역은 마이크로RNA 루프 서열 (예를 들면, miR-30a, miR-155, miR-218-1, 또는 miR-124-3 루프 서열 (예를 들면, 서열 번호: 758, 764, 767 또는 770 중 하나에서 선택된 핵산을 갖는 마이크로RNA 루프 서열)을 포함하고;
(iii) 표 2 또는 표 3에서 열거된 ASO 서열 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체) 중에서 하나에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과) 동일한 가이드 뉴클레오티드 서열을 포함하는 3' 스템 루프 팔을 포함하는 스템 루프 서열;
(b) 승객 가닥의 5'에 위치된 첫 번째 측부 영역 (예를 들면, 표 8에서 설명된 5' 측부 영역 중에서 하나); 그리고
(c) 가이드 가닥의 3'에 위치된 두 번째 측부 영역 (예를 들면, 표 8에서 설명된 3' 측부 영역 중에서 하나)으로서, 여기서 상기 두 번째 측부 영역은 3' 측부 서열 및 임의적으로, 3' 스페이서 서열을 포함한다.
다른 실례에서, 발현 벡터 또는 폴리뉴클레오티드는 다음을 인코딩하는 뉴클레오티드 서열을 포함하고:
(a) 스템 루프 서열로서, 5'에서 3'로:
(i) 표 2 또는 표 3에서 열거된 ASO 서열 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체) 중에서 하나에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과) 동일한 가이드 뉴클레오티드 서열을 포함하는 5' 스템 루프 팔;
(ii) 마이크로RNA 루프 영역으로서, 여기서 상기 루프 영역은 마이크로RNA 루프 서열 (예를 들면, miR-30a, miR-155, miR-218-1, 또는 miR-124-3 루프 서열 (예를 들면, 서열 번호: 758, 764, 767 또는 770 중 하나에서 선택된 핵산을 갖는 마이크로RNA 루프 서열)을 포함하고;
(iii) 가이드 뉴클레오티드 서열에 상보적이거나 또는 실질적으로 상보적인 승객 뉴클레오티드 서열을 포함하는 3' 스템 루프 팔을 포함하는 스템 루프 서열;
(b) 가이드 가닥의 5'에 위치된 5' 측부 영역 (예를 들면, 표 8에서 설명된 5' 측부 영역 중에서 하나); 그리고
(c) 승객 가닥의 3'에 위치된 3' 측부 영역 (예를 들면, 표 8에서 설명된 3' 측부 영역 중에서 하나)으로서, 여기서 상기 두 번째 측부 영역은 3' 측부 서열 및 임의적으로, 3' 스페이서 서열을 포함한다.
전술된 가이드 가닥 및 승객 가닥의 길이는 길이에서 19-50개 (예를 들면, 19, 20, 21, 22, 23, 24, 25, 26-30, 31-35, 36-40, 41-45, 또는 46-50개) 뉴클레오티드 사이일 수 있다. 특정 실례에서, 가이드 가닥의 길이는 19개 뉴클레오티드이다. 다른 실례에서, 가이드 가닥의 길이는 20개 뉴클레오티드이다. 다른 실례에서, 가이드 가닥의 길이는 21개 뉴클레오티드이다. 다른 실례에서, 가이드 가닥의 길이는 22개 뉴클레오티드이다. 다른 실례에서, 가이드 가닥의 길이는 23개 뉴클레오티드이다. 다른 실례에서, 가이드 가닥의 길이는 24개 뉴클레오티드이다. 다른 실례에서, 가이드 가닥의 길이는 25개 뉴클레오티드이다. 다른 실례에서, 가이드 가닥의 길이는 26-30개 뉴클레오티드이다. 다른 실례에서, 가이드 가닥의 길이는 31-35개 뉴클레오티드이다. 다른 실례에서, 가이드 가닥의 길이는 36-40개 뉴클레오티드이다. 다른 실례에서, 가이드 가닥의 길이는 41-45개 뉴클레오티드이다. 다른 실례에서, 가이드 가닥의 길이는 46-50개 뉴클레오티드이다. 특정 실례에서, 승객 가닥의 길이는 19개 뉴클레오티드이다. 다른 실례에서, 승객 가닥의 길이는 20개 뉴클레오티드이다. 다른 실례에서, 승객 가닥의 길이는 21개 뉴클레오티드이다. 다른 실례에서, 승객 가닥의 길이는 22개 뉴클레오티드이다. 다른 실례에서, 승객 가닥의 길이는 23개 뉴클레오티드이다. 다른 실례에서, 승객 가닥의 길이는 24개 뉴클레오티드이다. 다른 실례에서, 승객 가닥의 길이는 25개 뉴클레오티드이다. 다른 실례에서, 승객 가닥의 길이는 26-30개 뉴클레오티드이다. 다른 실례에서, 승객 가닥의 길이는 31-35개 뉴클레오티드이다. 다른 실례에서, 승객 가닥의 길이는 36-40개 뉴클레오티드이다. 다른 실례에서, 승객 가닥의 길이는 41-45개 뉴클레오티드이다. 다른 실례에서, 승객 가닥의 길이는 46-50개 뉴클레오티드이다.
가이드 및 승객 서열의 길이는 가이드 및 승객 가닥이 통합되는 miRNA 골격에 기초하여 변할 수 있다. 소정의 가이드가 miRNA 골격 내로 적응될 때, 가이드의 길이는 소정의 miRNA 골격의 자연 구조 및 처리를 수용하도록 확장될 수 있다. 예를 들면, E-miR-30 골격에 의해 생산된 가이드 서열은 전형적으로 22개 뉴클레오티드 길이이다. 대부분의 골격의 경우에, 가이드 서열은 표적 mRNA 서열에 추가적으로 상보적이도록 3' 단부에서 연장되지만, 일부 경우에는 miRNA 골격의 서열에 따라서, 가이드의 5' 시작 부위를 변형하는 것을 수반할 수 있다.
일정한 경우에, 소정의 작제물의 표적화 능력을 향상시키거나 또는 변경하기 위해 miRNA 가이드 및 승객 가닥 발현 수준 및/또는 처리 패턴을 변경하는 것이 바람직할 수 있다. 따라서, 소정의 miRNA 프레임워크/골격 내에, 가이드 및 승객 가닥의 위치는 교환될 수 있다 (도 6g); 이것은 스터퍼 서열을 포함하는 설계의 맥락에서 일 수 있거나, 또는 스터퍼가 없는 설계의 맥락에서 일 수 있다. 이것은 추가적으로 이중 작제물 (도 8g에서 도시된 바와 같음), 또는 연쇄체화된 작제물 (예컨대 도 8f)의 맥락에서 일 수 있다. 이러한 변화를 수용하기 위해, 가이드 및/또는 승객 가닥의 서열은 주형 "부모" 설계로부터 변형될 수 있다. 대안으로, 가이드 및 승객 가닥 발현 및/또는 처리 패턴에서 변화에 영향을 주기 위해, 가이드 및/또는 승객 가닥 서열에 변형이 만들어질 수 있다.
특정 실례에서, 벡터 또는 폴리뉴클레오티드는 첫 번째 측부 영역이 서열 번호: 752, 754, 756 및 759 중에서 하나에 적어도 90% (예를 들면, 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과) 동일한 뉴클레오티드 서열을 포함하는 miR-30a 서열을 포함한다 (표 8 참조).
일부 구체예에서, 벡터 또는 폴리뉴클레오티드는 두 번째 측부 영역이 서열 번호: 753, 755, 757 및 760 중에서 하나에 적어도 90% (예를 들면, 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과) 동일한 뉴클레오티드 서열을 포함하는 miR-30a 서열을 포함한다 (표 8 참조).
다른 실례에서, 벡터 또는 폴리뉴클레오티드는 루프 영역이 서열 번호: 758 또는 서열 번호: 761의 뉴클레오티드 서열, 또는 서열 번호: 758 또는 서열 번호: 761에 적어도 90% (예를 들면, 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과) 동일한 서열을 포함하는 miR-30a 구조를 포함한다 (표 8 참조).
특정 실례에서, 벡터 또는 폴리뉴클레오티드는 첫 번째 측부 영역이 서열 번호: 762에 적어도 90% (예를 들면, 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과) 동일한 뉴클레오티드 서열을 포함하는 miR-155 서열을 포함한다 (표 8 참조).
일부 구체예에서, 벡터 또는 폴리뉴클레오티드는 두 번째 측부 영역이 서열 번호: 763에 적어도 90% (예를 들면, 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과) 동일한 뉴클레오티드 서열을 포함하는 miR-155 서열을 포함한다 (표 8 참조).
다른 실례에서, 벡터 또는 폴리뉴클레오티드는 루프 영역이 서열 번호: 764의 뉴클레오티드 서열, 또는 서열 번호: 764에 적어도 90% (예를 들면, 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과) 동일한 서열을 포함하는 miR-155 구조를 포함한다 (표 8 참조).
특정 실례에서, 벡터 또는 폴리뉴클레오티드는 첫 번째 측부 영역이 서열 번호: 765에 적어도 90% (예를 들면, 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과) 동일한 뉴클레오티드 서열을 포함하는 miR-218-1 서열을 포함한다 (표 8 참조).
일부 구체예에서, 벡터 또는 폴리뉴클레오티드는 두 번째 측부 영역이 서열 번호: 766에 적어도 90% (예를 들면, 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과) 동일한 뉴클레오티드 서열을 포함하는 miR-218-1 서열을 포함한다 (표 8 참조).
다른 실례에서, 벡터 또는 폴리뉴클레오티드는 루프 영역이 서열 번호: 767의 뉴클레오티드 서열, 또는 서열 번호: 767에 적어도 90% (예를 들면, 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과) 동일한 서열을 포함하는 miR-218-1 구조를 포함한다 (표 8 참조).
특정 실례에서, 벡터 또는 폴리뉴클레오티드는 첫 번째 측부 영역이 서열 번호: 768에 적어도 90% (예를 들면, 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과) 동일한 뉴클레오티드 서열을 포함하는 miR-124-3 서열을 포함한다 (표 8 참조).
일부 구체예에서, 벡터 또는 폴리뉴클레오티드는 두 번째 측부 영역이 서열 번호: 769에 적어도 90% (예를 들면, 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과) 동일한 뉴클레오티드 서열을 포함하는 miR-124-3 서열을 포함한다 (표 8 참조).
다른 실례에서, 벡터 또는 폴리뉴클레오티드는 루프 영역이 서열 번호: 770의 뉴클레오티드 서열, 또는 서열 번호: 770에 적어도 90% (예를 들면, 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과) 동일한 서열을 포함하는 miR-124-3 구조를 포함한다 (표 8 참조).
발현 벡터는 플라스미드일 수 있고, 그리고 예를 들면, 인트론 서열 (예를 들면, 서열 번호: 743 또는 서열 번호: 744, 또는 서열 번호: 743 또는 서열 번호: 744의 핵산 서열에 적어도 85% (예를 들면, 적어도 85% 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체의 인트론 서열), 링커 서열, 또는 스터퍼 서열 중에서 하나 이상을 포함할 수 있다.
표 8. 마이크로RNA 서열
따라서, 본원 발명의 목적은 서열 번호: 1-100 중에서 하나의 핵산 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 폴리뉴클레오티드를 포함하는 벡터에 관계한다. 예를 들면, 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 90% (예를 들면, 적어도 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 폴리뉴클레오티드를 포함할 수 있다. 다른 실례에서, 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 95% (예를 들면, 적어도 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 폴리뉴클레오티드를 포함할 수 있다. 본원 발명의 벡터는 서열 번호: 1-100 중에서 하나의 핵산 서열을 갖는 폴리뉴클레오티드를 더욱 포함할 수 있다.
특히, 벡터는 서열 번호: 1-100 중에서 하나의 서열 또는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체 및 프로모터 (예를 들면, 표 5 또는 표 6, 또는 a에서 열거된 프로모터 중에서 하나)를 포함할 수 있다.
상기 논의된 변이체는 예를 들면, 개체 사이에 대립유전자 변동 (예를 들면, 다형성), 대안적 스플라이싱 형태 등에 기인한 자연 발생 변이체를 포함할 수 있다. 용어 변이체는 또한, 다른 공급원 또는 생물체로부터 본원 발명의 유전자 서열을 포함한다. 변이체는 본원 발명에 따른 서열과 실제적으로 상동할 수 있다. 본원 발명의 유전자의 변이체는 또한, 엄격한 혼성화 조건하에 앞서 규정된 바와 같은 서열 (또는 이의 상보성 가닥)에 혼성화하는 핵산 서열을 포함한다. 전형적인 엄격한 혼성화 조건은 30℃ 초과, 35℃ 초과, 또는 42℃ 초과의 온도 및/또는 약 500 mM 미만 또는 200 mM 미만의 염도를 포함한다. 혼성화 조건은 예를 들면, 온도, 염도 및/또는 다른 시약 예컨대 SDS, SSC 등의 농도를 변경함으로써 조정될 수 있다.
본원 발명은 이종성 폴리뉴클레오티드의 관심되는 표적 세포로의 전달을 위한 비바이러스 벡터 (예를 들면, 본원에서 개시된 Grik2 표적화 ASO 작용제를 인코딩하는 폴리뉴클레오티드를 내포하는 플라스미드)를 더욱 제공한다. 다른 사례에서, 본원 발명의 바이러스 벡터는 AAV 벡터 아데노바이러스, 레트로바이러스, 렌티바이러스, 또는 헤르페스바이러스 벡터일 수 있다.
하나 이상의 발현 카세트가 이용될 수 있다. 각 발현 카세트는 관심되는 RNA를 인코딩하는 서열에 작동가능하게 연결된 프로모터 서열 (예를 들면, 뉴런 세포 프로모터)을 적어도 포함할 수 있다. 각 발현 카세트는 추가의 조절 요소, 스페이서, 인트론, UTR, 폴리아데닐화 부위 등으로 구성될 수 있다. 발현 카세트는 예를 들면 2개 이상의 ASO 작용제를 인코딩하는 폴리뉴클레오티드에 대하여 다중유전자일 수 있다. 발현 카세트는 프로모터, 관심되는 하나 이상의 ASO 작용제를 인코딩하는 핵산 및 폴리A 서열을 더욱 포함할 수 있다. 특정 실례에서, 발현 카세트는 5' - 프로모터 서열, 관심되는 첫 번째 ASO 작용제 (예를 들면, 서열 번호: 1-100 중에서 하나)를 인코딩하는 폴리뉴클레오티드 서열, 관심되는 두 번째 ASO 작용제 (예를 들면, 서열 번호: 1-100 중에서 하나)를 인코딩하는 서열, 그리고 폴리A 서열- 3'을 포함한다.
바이러스 벡터는 항생제 내성 유전자 예컨대 내성 AmpR, 카나마이신, 히그로마이신 B, 제네티신, 블라스티시딘 S, 젠타마이신, 카르베니실린, 클로람페니콜, 누르세오트리신, 또는 푸로마이신의 유전자를 인코딩하는 핵산 서열을 더욱 포함할 수 있다.
예시적인 발현 카세트
본원 발명은, 발현 벡터 (예를 들면, 플라스미드 또는 바이러스 벡터 (예를 들면, AAV 또는 렌티바이러스 벡터)) 내로 통합될 때, Grik2 mRNA에 혼성화하고 이것의 발현을 저해하는 ASO 작용제 (예를 들면, 서열 번호: 1-100 중에서 하나의 핵산 서열을 갖는 ASO 작용제)를 인코딩하는 이종성 폴리뉴클레오티드의 발현을 증진하는 발현 카세트를 제공한다. 일반적으로, 핵산 벡터 내로 통합된 발현 카세트는 이종성 유전자 조절 서열 (예를 들면, 프로모터 (예를 들면, 표 5 또는 표 6에서 설명된 프로모터 중에서 하나) 및 임의적으로, 인핸서 서열 (예를 들면, 표 7에서 설명된 인핸서 서열)), 5' 측부 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나), 스템 루프 5' 팔, 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 또는 770 중에서 하나) 및 스템 루프 3' 팔을 내포하는 스템 루프 서열, 3' 측부 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 또는 769 중에서 하나), 임의적으로, 우드척 간염 전사후 조절 요소 (WRPE), 그리고 폴리A 서열 (예를 들면, 서열 번호: 750, 751, 792 또는 793)을 내포하는 이종성 폴리뉴클레오티드를 포함할 것이다. AAV 벡터의 경우에, 발현 카세트는 자체의 5'와 3' 단부에서 각각, 5' ITR 및 3' ITR 서열 (예를 들면, 표 7에서 설명된 5' 또는 3' ITR 서열 중에서 하나)과 측접될 수 있다. 전형적으로, AAV2 ITR 서열이 본원에서 개시된 방법과 조성물용으로 예기되지만, 본원에서 개시된 다른 AAV 혈청형으로부터 유래된 ITR 서열이 또한 이용될 수 있다 (상기 섹션 "AAV 벡터"를 참조한다). 본원 발명의 범위를 제한하지 않고, 그리고 개시된 방법 및 조성물용으로 적합한 발현 카세트를 단지 예시하기 위해, U.S. 특허가출원 번호: 63/050,742로부터 본원에서 온전히 참조로서 편입되는 표 9 및 표 10은 각각, 도입유전자 발현을 뉴런에서 또는 편재성으로 유도하는 데 유용한, 5'에서 3' 방향으로 이동하는 발현 카세트 요소를 갖는 예시적인 발현 카세트 작제물을 특징으로 한다. 작제물의 일반적인 구조는 5'에서 3' 방향으로 배향된 하기 요소를 적어도 포함한다:
(i) 5' ITR 서열 (AAV 벡터 단독의 경우; 예를 들면, 서열 번호: 746 또는 서열 번호: 747);
(ii) 프로모터 서열 (예를 들면, 표 5 또는 표 6에서 열거된 프로모터 서열 중에서 하나);
(iii) 5' 측부 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나);
(iv) 5'에서 3' 방향으로, 다음을 포함하는 스템 루프 서열:
a. 스템 루프 5' 팔로서, 여기서 상기 스템 루프 5' 팔은 서열 번호: 1-100 중에서 하나의 ASO 서열 중에서 하나의 ASO 서열을 적어도 내포하는 가이드 서열, 또는 서열 번호: 1-100에 상보적이거나 또는 실제적으로 상보적인 (예를 들면, 10개 이내, 9개 이내, 8개 이내, 7개 이내, 6개 이내, 5개 이내, 4개 이내, 3개 이내, 2개 이내, 또는 1개 이내의 부정합된 뉴클레오티드를 내포하는) 승객 서열을 포함하고;
b. 루프 서열 (예를 들면, miR-30, miR-155, miR-218-1, 또는 miR-124-3 루프 서열, 예컨대 예를 들면, 서열 번호: 758, 761, 764, 767 또는 770 중에서 하나의 루프 서열); 그리고
c. 스템 루프 3' 팔로서, 여기서 상기 스템 루프 3' 팔은 서열 번호: 1-100 중에서 하나의 ASO 서열을 적어도 내포하는 가이드 서열, 또는 서열 번호: 1-100 중에서 하나의 ASO 서열에 실제적으로 상보적인 (예를 들면, 10개 이내, 9개 이내, 8개 이내, 7개 이내, 6개 이내, 5개 이내, 4개 이내, 3개 이내, 2개 이내, 또는 1개 이내의 부정합된 뉴클레오티드를 내포하는) 승객 서열을 포함하고;
(v) 3' 측부 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 또는 769 중에서 하나);
(vi) 임의적으로, WPRE 서열;
(vii) 폴리A 서열 (예를 들면, 서열 번호: 750, 서열 번호: 751, 서열 번호: 792, 또는 서열 번호: 793); 그리고
(viii) 3' ITR 서열 (AAV 벡터 단독의 경우; 예를 들면, 서열 번호: 748, SEQ ID O: 749, 또는 서열 번호: 789).
특정 실례에서, 본원 발명은 표 4에서 설명된 표적 서열로 구성된 군에서 선택되는 Grik2 mRNA 표적 서열 또는 서열 번호: 164-681 중에서 하나, 또는 표 4에서 설명된 상응하는 표적 서열 또는 서열 번호: 164-681 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에 완전히 또는 실제적으로 상보적인 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나)를 포함하는 발현 카세트를 제공한다.
다른 실례에서, 본원 발명은 표 4에서 설명된 표적 서열로 구성된 군에서 선택되는 Grik2 mRNA 표적 서열 또는 서열 번호: 164-681 중에서 하나, 또는 표 4에서 설명된 상응하는 표적 서열 또는 서열 번호: 164-681 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에 완전히 또는 실제적으로 상보적인 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나)를 포함하는 발현 카세트를 제공한다.
다른 실례에서, 본원 발명은 표 4에서 설명된 표적 서열로 구성된 군에서 선택되는 Grik2 mRNA 표적 서열 또는 서열 번호: 164-681 중에서 하나, 또는 표 4에서 설명된 상응하는 표적 서열 또는 서열 번호: 164-681 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에 완전히 또는 실제적으로 상보적인 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 CAG 프로모터 (예를 들면, 서열 번호: 737)를 포함하는 발현 카세트를 제공한다.
다른 실례에서, 본원 발명은 표 4에서 설명된 표적 서열로 구성된 군에서 선택되는 Grik2 mRNA 표적 서열 또는 서열 번호: 164-681 중에서 하나, 또는 표 4에서 설명된 상응하는 표적 서열 또는 서열 번호: 164-681 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에 완전히 또는 실제적으로 상보적인 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 CBA 프로모터 (예를 들면, 서열 번호: 738)를 포함하는 발현 카세트를 제공한다.
다른 실례에서, 본원 발명은 표 4에서 설명된 표적 서열로 구성된 군에서 선택되는 Grik2 mRNA 표적 서열 또는 서열 번호: 164-681 중에서 하나, 또는 표 4에서 설명된 상응하는 표적 서열 또는 서열 번호: 164-681 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에 완전히 또는 실제적으로 상보적인 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 U6 프로모터 (예를 들면, 서열 번호: 728-733 중에서 하나)를 포함하는 발현 카세트를 제공한다.
다른 실례에서, 본원 발명은 표 4에서 설명된 표적 서열로 구성된 군에서 선택되는 Grik2 mRNA 표적 서열 또는 서열 번호: 164-681 중에서 하나, 또는 표 4에서 설명된 상응하는 표적 서열 또는 서열 번호: 164-681 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에 완전히 또는 실제적으로 상보적인 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 H1 프로모터 (예를 들면, 서열 번호: 734)를 포함하는 발현 카세트를 제공한다.
다른 실례에서, 본원 발명은 표 4에서 설명된 표적 서열로 구성된 군에서 선택되는 Grik2 mRNA 표적 서열 또는 서열 번호: 164-681 중에서 하나, 또는 표 4에서 설명된 상응하는 표적 서열 또는 서열 번호: 164-681 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에 완전히 또는 실제적으로 상보적인 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 7SK 프로모터 (예를 들면, 서열 번호: 735)를 포함하는 발현 카세트를 제공한다.
다른 실례에서, 본원 발명은 서열 번호: 1-100 중에서 하나, 또는 서열 번호: 1-100 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체로 구성된 군에서 선택되는 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나)를 포함하는 발현 카세트를 제공한다.
다른 실례에서, 본원 발명은 서열 번호: 1-100 중에서 하나, 또는 서열 번호: 1-100 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체로 구성된 군에서 선택되는 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나)를 포함하는 발현 카세트를 제공한다.
다른 실례에서, 본원 발명은 서열 번호: 1-100 중에서 하나, 또는 서열 번호: 1-100 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체로 구성된 군에서 선택되는 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 CAG 프로모터 (예를 들면, 서열 번호: 737)를 포함하는 발현 카세트를 제공한다.
다른 실례에서, 본원 발명은 서열 번호: 1-100 중에서 하나, 또는 서열 번호: 1-100 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체로 구성된 군에서 선택되는 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 CBA 프로모터 (예를 들면, 서열 번호: 738)를 포함하는 발현 카세트를 제공한다.
다른 실례에서, 본원 발명은 서열 번호: 1-100 중에서 하나, 또는 서열 번호: 1-100 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체로 구성된 군에서 선택되는 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 U6 프로모터 (예를 들면, 서열 번호: 728-733 중에서 하나)를 포함하는 발현 카세트를 제공한다.
다른 실례에서, 본원 발명은 서열 번호: 1-100 중에서 하나, 또는 서열 번호: 1-100 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체로 구성된 군에서 선택되는 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 H1 프로모터 (예를 들면, 서열 번호: 734)를 포함하는 발현 카세트를 제공한다.
다른 실례에서, 본원 발명은 서열 번호: 1-100 중에서 하나, 또는 서열 번호: 1-100 중에서 하나에 적어도 85% (적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체로 구성된 군에서 선택되는 항-Grik2 가이드 서열 및 상기 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열을 포함하는 폴리뉴클레오티드에 작동가능하게 연결된 7SK 프로모터 (예를 들면, 서열 번호: 735)를 포함하는 발현 카세트를 제공한다.
다른 실례에서, 본원 발명은 아래의 표 9에서 설명된 발현 카세트 중 하나에서 선택되는 발현 카세트를 제공한다.
표 9: 예시적인 발현 카세트
표 9 핵심:
A = 서열 번호: 746 및 747에서 선택되는 AAV 5' ITR 서열;
B = 서열 번호: 752, 754, 756, 759, 762, 765 및 768로 구성된 군에서 선택되는 스템 루프 5' 측부 서열;
C = 서열 번호: 758, 761, 764, 767 및 770으로 구성된 군에서 선택되는 마이크로RNA 루프 서열;
D = 서열 번호: 753, 754, 757, 760, 763, 766 및 769로 구성된 군에서 선택되는 스템 루프 3' 측부 서열;
E = 서열 번호: 750 및 751에서 선택되는 폴리뉴클레오티드를 내포하는 3' 비번역 영역 (UTR);
F = 서열 번호: 748 및 749에서 선택되는 AAV 3' ITR 서열.
다른 실례에서, 서열 번호: 1-100 중에서 하나의 ASO 서열에 실제적으로 상보적인 승객 서열은 서열 번호: 1-100 중에서 하나의 ASO 서열에 비하여 5개 이내 (예를 들면, 5, 4, 3, 2, 또는 1개 이내)의 부정합된 뉴클레오티드 (다시 말하면, 부정합)을 갖는다. 다른 실례에서, 서열 번호: 1-100 중에서 하나의 ASO 서열에 실제적으로 상보적인 승객 서열은 서열 번호: 1-100 중에서 하나의 ASO 서열에 비하여 4개 이내 (예를 들면, 4, 3, 2, 또는 1개 이내)의 부정합을 갖는다. 다른 실례에서, 서열 번호: 1-100 중에서 하나의 ASO 서열에 실제적으로 상보적인 승객 서열은 서열 번호: 1-100 중에서 하나의 ASO 서열에 비하여 3개 이내 (예를 들면, 3, 2, 또는 1개 이내)의 부정합을 갖는다. 다른 실례에서, 서열 번호: 1-100 중에서 하나의 ASO 서열에 실제적으로 상보적인 승객 서열은 서열 번호: 1-100 중에서 하나의 ASO 서열에 비하여 2개 이내 (예를 들면, 2 또는 1개 이내)의 부정합을 갖는다. 또 다른 실례에서, 서열 번호: 1-100 중에서 하나의 ASO 서열에 실제적으로 상보적인 승객 서열은 서열 번호: 1-100 중에서 하나의 ASO 서열에 비하여 1개 이내의 부정합을 갖는다.
다른 실례에서, 서열 번호: 1-100 중에서 하나의 ASO 서열에 실제적으로 상보적인 승객 서열은 서열 번호: 1-100 중에서 하나의 ASO 서열에 비하여 10개 이내 (예를 들면, 10, 9, 8, 7, 또는 6개 이내)의 부정합된 뉴클레오티드 (다시 말하면, 부정합)을 갖는다. 다른 실례에서, 서열 번호: 1-100 중에서 하나의 ASO 서열에 실제적으로 상보적인 승객 서열은 서열 번호: 1-100 중에서 하나의 ASO 서열에 비하여 9개 이내 (예를 들면, 9, 8, 7, 또는 6개 이내)의 부정합을 갖는다. 다른 실례에서, 서열 번호: 1-100 중에서 하나의 ASO 서열에 실제적으로 상보적인 승객 서열은 서열 번호: 1-100 중에서 하나의 ASO 서열에 비하여 8개 이내 (예를 들면, 8, 7, 또는 6개 이내)의 부정합을 갖는다. 다른 실례에서, 서열 번호: 1-100 중에서 하나의 ASO 서열에 실제적으로 상보적인 승객 서열은 서열 번호: 1-100 중에서 하나의 ASO 서열에 비하여 7개 이내 (예를 들면, 7 또는 6개 이내)의 부정합을 갖는다. 다른 실례에서, 서열 번호: 1-100 중에서 하나의 ASO 서열에 실제적으로 상보적인 승객 서열은 서열 번호: 1-100 중에서 하나의 ASO 서열에 비하여 6개 이내의 부정합을 갖는다. 다른 실례에서, 서열 번호: 1-100 중에서 하나의 ASO 서열에 실제적으로 상보적인 승객 서열은 서열 번호: 1-100 중에서 하나의 ASO 서열에 비하여 5개 이내의 부정합을 갖는다. 다른 실례에서, 서열 번호: 1-100 중에서 하나의 ASO 서열에 실제적으로 상보적인 승객 서열은 서열 번호: 1-100 중에서 하나의 ASO 서열에 비하여 4개 이내의 부정합을 갖는다. 다른 실례에서, 서열 번호: 1-100 중에서 하나의 ASO 서열에 실제적으로 상보적인 승객 서열은 서열 번호: 1-100 중에서 하나의 ASO 서열에 비하여 3개 이내의 부정합을 갖는다. 다른 실례에서, 서열 번호: 1-100 중에서 하나의 ASO 서열에 실제적으로 상보적인 승객 서열은 서열 번호: 1-100 중에서 하나의 ASO 서열에 비하여 2개 이내의 부정합을 갖는다. 다른 실례에서, 서열 번호: 1-100 중에서 하나의 ASO 서열에 실제적으로 상보적인 승객 서열은 서열 번호: 1-100 중에서 하나의 ASO 서열에 비하여 1개 이내의 부정합을 갖는다.
본원에서 온전히 참조로서 편입되는 U.S. 특허가출원 번호: 63/050,742의 표 9 또는 표 10에서 예시된 발현 작제물은 추가의 벡터 요소 예컨대 예를 들면, 조절 서열 (예를 들면, 하나 이상의 인핸서 서열, 종결자 서열, 또는 WPRE 서열), 스터퍼 및 임의의 설명된 요소 사이에 또는 내에 링커 서열뿐만 아니라 세포에서 이종성 폴리뉴클레오티드의 발현을 증진하는 데 이용될 수 있는 당해 분야에서 공지된 임의의 다른 전통적인 발현 작제물 요소를 더욱 포함할 수 있다. 표 9 및 표 10은 예시적인 발현 카세트를 제공하는데, 이들은 각각 단일 열 내에 도시되고 식별자 번호 (예를 들면, 표 9의 예시적인 발현 카세트 입체형상 1-3800 및 표 10의 입체형상 1-2000)에 의해 지정되며, 그리고 발현 카세트의 각 요소는 5'에서 3' 방향으로 정향된 일련의 칼럼에 나타내진다.
본원 발명의 예시적인 단일시스트론성 (다시 말하면, 단일 ASO-인코딩) 작제물은 hSyn 프로모터 (서열 번호: 790) 및 A-miR-30 골격 내로 통합된 ASO G9 (서열 번호: 68)를 내포하는 AAV (예를 들면, AAV9) 작제물을 포함할 수 있다 (작제물 1; 도 6a 참조). 이런 작제물은 서열 번호: 775의 핵산 서열을 가질 수 있거나, 또는 서열 번호: 775의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다 (하기 참조).
(서열 번호: 775)
핵심: 굵은 글씨 = 5' ITR 서열; 단일 밑줄 = 프로모터 서열; 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+굵은 글씨+단일 밑줄 : G9 가이드 서열을 인코딩하는 DNA; 이탤릭체+물결선 밑줄: G9 승객 서열을 인코딩하는 DNA; 이중 밑줄: 폴리A 서열; 굵은 글씨+소문자: 3' ITR 서열 (서열 번호: 789).
전술된 예시적인 단일시스트론성, 항-Grik2 작제물은 마이크로RNA 코딩 서열이 서열 번호: 795의 핵산 서열을 갖는 폴리뉴클레오티드이거나, 또는 서열 번호: 795의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이도록, A-miR-30 골격 내로 통합된 Grik2 안티센스 가이드 서열 (예를 들면, G9, 서열 번호: 68)을 포함할 수 있다.
(서열 번호: 795)
핵심: 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+물결선 밑줄: 승객 서열을 인코딩하는 DNA; 이탤릭체+굵은 글씨+단일 밑줄 : G9 가이드 서열을 인코딩하는 DNA.
본원 발명의 다른 예시적인 단일시스트론성, 항-Grik2 작제물은 탠덤으로 C1ql2 프로모터 (서열 번호: 791) 및 hSyn 프로모터 (서열 번호: 790), 그리고 A-miR-30 골격 내로 통합된 ASO G9 (서열 번호: 68)를 내포하는 AAV (예를 들면, AAV9) 작제물을 포함할 수 있다 (작제물 2; 도 6b 참조). 이런 작제물은 서열 번호: 777의 핵산 서열을 가질 수 있거나, 또는 서열 번호: 777의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다 (하기 참조).
(서열 번호: 777)
핵심: 굵은 글씨 = 5' ITR 서열; 단일 밑줄+이탤릭체 = C1ql2 프로모터 서열; 단일 밑줄: hSyn 프로모터 서열; 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+물결선 밑줄: 승객 서열을 인코딩하는 DNA; 이탤릭체+굵은 글씨+단일 밑줄 : G9 가이드 서열을 인코딩하는 DNA; 이중 밑줄: 폴리A 서열; 굵은 글씨+소문자: 3' ITR 서열 (서열 번호: 789).
전술된 예시적인 단일시스트론성, 항-Grik2 작제물은 마이크로RNA 코딩 서열이 서열 번호: 778의 핵산 서열을 갖는 폴리뉴클레오티드이거나, 또는 서열 번호: 778의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이도록, 마이크로RNA 골격 내로 통합된 Grik2 안티센스 가이드 서열 (예를 들면, G9, 서열 번호: 68)을 포함할 수 있다.
(서열 번호: 778)
핵심: 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+물결선 밑줄: 승객 서열을 인코딩하는 DNA; 이탤릭체+굵은 글씨+단일 밑줄 : G9 가이드 서열을 인코딩하는 DNA.
본원 발명의 다른 예시적인 단일시스트론성, 항-Grik2 작제물은 5' ITR 서열의 하류에 hSyn 프로모터 (서열 번호: 790) 및 A-miR-30 골격 내로 통합된 ASO G9 (서열 번호: 68)를 내포하는 자가 상보성 (sc)AAV (예를 들면, scAAV9) 작제물을 포함할 수 있다 (작제물 3; 도 6c 참조). 이런 작제물은 서열 번호: 779의 핵산 서열을 가질 수 있거나, 또는 서열 번호: 779의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다 (하기 참조).
(서열 번호: 779)
핵심: 굵은 글씨 = 5' ITR 서열; 단일 밑줄+이탤릭체 = hSyn 프로모터 서열; 단일 밑줄: hSyn 프로모터 서열; 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+물결선 밑줄: 승객 서열을 인코딩하는 DNA; 이탤릭체+굵은 글씨+단일 밑줄 : G9 가이드 서열을 인코딩하는 DNA; 이중 밑줄: RBG 폴리A 서열; 이중 밑줄+굵은 글씨 = BGH 폴리A 서열 (서열 번호: 793); 굵은 글씨+소문자: 3' ITR 서열 (서열 번호: 748).
전술된 예시적인 단일시스트론성, 항-Grik2 작제물은 마이크로RNA 코딩 서열이 서열 번호: 780의 핵산 서열을 갖는 폴리뉴클레오티드이거나, 또는 서열 번호: 780의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이도록, 마이크로RNA 골격 내로 통합된 Grik2 안티센스 가이드 서열 (예를 들면, G9, 서열 번호: 68)을 포함할 수 있다.
(서열 번호: 780)
핵심: 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+물결선 밑줄: 승객 서열을 인코딩하는 DNA; 이탤릭체+굵은 글씨+ 단일 밑줄 : G9 가이드 서열을 인코딩하는 DNA.
본원 발명의 다른 예시적인 단일시스트론성, 항-Grik2 작제물은 3' ITR의 근위에 hSyn 프로모터 (서열 번호: 790) ("FLIP") 및 A-miR-30 골격 내로 통합된 ASO G9 (서열 번호: 68)를 내포하는 scAAV (예를 들면, scAAV9) 작제물을 포함할 수 있다 (작제물 4; 도 6d 참조). 이런 작제물은 서열 번호: 781의 핵산 서열을 가질 수 있거나, 또는 서열 번호: 781의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다 (하기 참조).
(서열 번호: 781)
핵심: 굵은 글씨 = 5' ITR 서열; 단일 밑줄+이탤릭체 = hSyn 프로모터 서열; 단일 밑줄: hSyn 프로모터 서열; 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+물결선 밑줄: 승객 서열을 인코딩하는 DNA; 이탤릭체+굵은 글씨+단일 밑줄 : G9 가이드 서열을 인코딩하는 DNA; 이중 밑줄: RBG 폴리A 서열; 이중 밑줄+굵은 글씨 = BGH 폴리A 서열 (서열 번호: 793); 굵은 글씨+소문자: 3' ITR 서열 (서열 번호: 748). 전체 카세트가 5' ITR에서 3' ITR 대신에 3' ITR에서 5'ITR로 판독되기 때문에, 이 경우에 있어서 가이드 서열이 "역방향"이라는 점에 주의한다.
전술된 예시적인 단일시스트론성, 항-Grik2 작제물은 마이크로RNA 코딩 서열이 서열 번호: 782 (위쪽 가닥) 또는 서열 번호: 794 (아래쪽 가닥)의 핵산 서열을 갖는 폴리뉴클레오티드이거나, 또는 서열 번호: 782 또는 서열 번호: 794의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이도록, 마이크로RNA 골격 내로 통합된 Grik2 안티센스 가이드 서열 (예를 들면, G9, 서열 번호: 68)을 포함할 수 있다.
(서열 번호: 782)
(서열 번호: 794)
핵심: 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+물결선 밑줄: 승객 서열을 인코딩하는 DNA; 이탤릭체+굵은 글씨+단일 밑줄 : G9 가이드 서열을 인코딩하는 DNA.
본원 발명의 다른 예시적인 단일시스트론성, 항-Grik2 작제물은 hSyn 프로모터 (서열 번호: 790) 및 A-miR-30 골격 내로 통합된 ASO G9 (서열 번호: 68)의 3개의 연쇄체화된 사본을 내포하는 AAV (예를 들면, AAV9) 작제물을 포함할 수 있다 (작제물 5; 도 6e 참조). 이런 작제물은 서열 번호: 783의 핵산 서열을 가질 수 있거나, 또는 서열 번호: 783의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다 (하기 참조).
(서열 번호: 783)
핵심: 굵은 글씨 = 5' ITR 서열; 단일 밑줄 = 프로모터 서열; 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+물결선 밑줄: 승객 서열을 인코딩하는 DNA; 이탤릭체+굵은 글씨+단일 밑줄 : G9 가이드 서열을 인코딩하는 DNA; 이중 밑줄: 폴리A 서열; 굵은 글씨 + 소문자: 3' ITR 서열 (서열 번호: 789); ^ = 첫 번째 연쇄체의 경계; * = 두 번째 연쇄체의 경계; 및 # = 세 번째 연쇄체의 경계.
본원 발명의 다른 예시적인 단일시스트론성, 항-Grik2 작제물은 hSyn 프로모터 (서열 번호: 790) 및 A-miR-30 골격 내로 각각 통합된 G9 (서열 번호: 68), GI (서열 번호: 77), MU (서열 번호: 96)를 포함하는 상이한 안티센스 서열의 3개의 연쇄체화된 사본을 내포하는 AAV (예를 들면, AAV9) 작제물을 포함할 수 있다 (작제물 6; 도 6e 참조). 이런 작제물은 서열 번호: 784의 핵산 서열을 가질 수 있거나, 또는 서열 번호: 784의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다 (하기 참조).
(서열 번호: 784)
핵심: 굵은 글씨 = 5' ITR 서열; 단일 밑줄 = 프로모터 서열; 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+물결선 밑줄: 가이드 서열 (G9, GI, MU - 상기 순서로)에 상보적인 승객 서열을 인코딩하는 DNA; 이탤릭체+굵은 글씨+단일 밑줄 : 가이드 서열 (G9, GI, MU - 상기 순서로)을 인코딩하는 DNA; 이중 밑줄: 폴리A 서열; 굵은 글씨+소문자: 3' ITR 서열 (서열 번호: 789); ^ = 첫 번째 연쇄체의 경계; * = 두 번째 연쇄체의 경계; 및 # = 세 번째 연쇄체의 경계.
다른 사례에서, G9 ASO 서열 (서열 번호: 68)은 마이크로RNA 코딩 서열이 서열 번호: 801의 핵산 서열을 갖는 폴리뉴클레오티드이거나, 또는 서열 번호: 801의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이도록, E-miR-124-3 골격 내로 통합될 수 있다.
(서열 번호: 801)
핵심: 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+물결선 밑줄: G9 승객 서열을 인코딩하는 DNA 이탤릭체+굵은 글씨+단일 밑줄 : G9 가이드 서열을 인코딩하는 DNA.
본원 발명의 다른 단일시스트론성, 항-Grik2 작제물은 hSyn 프로모터 (서열 번호: 790) 및 E-miR-124-3 골격 내로 통합된 항-Grik2 안티센스 서열 G9 (서열 번호: 68)를 내포하는 AAV (예를 들면, AAV9) 작제물이다. 이런 작제물은 서열 번호: 809의 핵산 서열을 가질 수 있거나, 또는 서열 번호: 809의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다 (하기 참조). 서열 번호: 809의 발현 작제물은 서열 번호: 810의 핵산 서열 또는 서열 번호: 810의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 벡터 내로 통합될 수 있다.
(서열 번호: 809)
핵심: 단일 밑줄: 프로모터 서열; 굵은 글씨: 가이드 및 승객 서열을 내포하는 마이크로RNA 스템 루프 구조; 이중 밑줄: 폴리A 서열; 이탤릭체: 스터퍼 서열 1; 이탤릭체+밑줄 : 스터퍼 서열 2.
본원 발명의 다른 단일시스트론성, 항-Grik2 작제물은 hSyn 프로모터 (서열 번호: 790) 및 A-miR-30 골격 내로 통합된 ASO GI (서열 번호: 77)를 내포하는 AAV (예를 들면, AAV9) 작제물이다. 이런 작제물은 서열 번호: 796의 핵산 서열을 가질 수 있거나, 또는 서열 번호: 817의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다 (하기 참조).
(서열 번호: 817)
핵심: 단일 밑줄 = 프로모터 서열; 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+물결선 밑줄: GI 승객 서열을 인코딩하는 DNA; 이탤릭체+굵은 글씨+단일 밑줄 : GI 가이드 서열을 인코딩하는 DNA 이중 밑줄: 폴리A 서열.
서열 번호: 817의 작제물은 아래의 서열 번호: 796에 나타나 있는 바와 같이, 5'와 3' ITR 서열을 더욱 내포하는 벡터 내로 통합될 수 있다.
(서열 번호: 796)
핵심: 굵은 글씨 = 5' ITR 서열; 단일 밑줄 = 프로모터 서열; 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+물결선 밑줄: GI 승객 서열을 인코딩하는 DNA; 이탤릭체+굵은 글씨+단일 밑줄 : GI 가이드 서열을 인코딩하는 DNA 이중 밑줄: 폴리A 서열; 굵은 글씨+소문자: 3' ITR 서열 (서열 번호: 789).
전술된 예시적인 단일시스트론성, 항-Grik2 작제물은 마이크로RNA 코딩 서열이 서열 번호: 797의 핵산 서열을 갖는 폴리뉴클레오티드이거나, 또는 서열 번호: 797의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이도록, A-miR-30 골격 내로 통합된 Grik2 안티센스 가이드 서열 (예를 들면, GI, 서열 번호: 77)을 포함할 수 있다.
(서열 번호: 797)
핵심: 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+물결선 밑줄: GI 승객 서열을 인코딩하는 DNA; 이탤릭체+굵은 글씨+밑줄 : GI 가이드 서열을 인코딩하는 DNA.
GI 항-Grik2 서열 (서열 번호: 77)을 통합하는 다른 항-Grik2 작제물은 마이크로RNA 코딩 서열이 서열 번호: 798의 핵산 서열을 갖는 폴리뉴클레오티드이거나, 또는 서열 번호: 798의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이도록, E-miR-30 골격을 포함할 수 있다.
(서열 번호: 798)
핵심: 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+굵은 글씨+단일 밑줄 : GI 가이드 서열을 인코딩하는 DNA; 이탤릭체+물결선 밑줄: GI 승객 서열을 인코딩하는 DNA.
본원 발명의 다른 단일시스트론성, 항-Grik2 작제물은 hSyn 프로모터 (서열 번호: 790), E-miR-30 골격 내로 통합된 ASO GI (서열 번호: 77), 그리고 스터퍼 서열 (서열 번호: 815 및 816)을 내포하는 AAV (예를 들면, AAV9) 작제물이다. 이런 작제물은 서열 번호: 803의 핵산 서열을 가질 수 있거나, 또는 서열 번호: 803의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다 (하기 참조). 서열 번호: 803 또는 이의 변이체의 발현 작제물은 서열 번호: 804의 핵산 서열 또는 서열 번호: 804의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 벡터 내로 통합될 수 있다.
(서열 번호: 803)
핵심: 단일 밑줄: 프로모터 서열; 굵은 글씨: 가이드 및 승객 서열을 내포하는 마이크로RNA 스템 루프 구조; 이중 밑줄: 폴리A 서열; 이탤릭체: 스터퍼 서열 1 (서열 번호: 815); 이탤릭체+밑줄 : 스터퍼 서열 2 (서열 번호: 816).
안티센스 작제물이 ASO 서열 MW (서열 번호: 80)를 내포하는 경우에, 상기 작제물은 마이크로RNA 코딩 서열이 서열 번호: 799의 핵산 서열을 갖는 폴리뉴클레오티드이거나, 또는 서열 번호: 799의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이도록, E-miR-218-1 골격을 포함할 수 있다.
(서열 번호: 799)
핵심: 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+굵은 글씨+단일 밑줄 : GI 가이드 서열을 인코딩하는 DNA; 이탤릭체+물결선 밑줄: GI 승객 서열을 인코딩하는 DNA.
서열 번호: 799의 작제물은 아래의 서열 번호: 819에 나타나 있는 바와 같이, hSyn 프로모터 (서열 번호: 790) 및 폴리A 서열을 더욱 포함할 수 있다.
(서열 번호: 819)
핵심: 단일 밑줄 = 프로모터 서열; 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+물결선 밑줄: GI 승객 서열을 인코딩하는 DNA; 이탤릭체+굵은 글씨+단일 밑줄 : GI 가이드 서열을 인코딩하는 DNA 이중 밑줄: 폴리A 서열.
본원 발명의 다른 단일시스트론성, 항-Grik2 작제물은 hSyn 프로모터 (서열 번호: 790), E-miR-218-1 골격 내로 통합된 ASO MW (서열 번호: 80), 그리고 하나 이상의 스터퍼 서열 (예를 들면, 서열 번호: 815 및/또는 서열 번호: 816)을 내포하는 AAV (예를 들면, AAV9) 작제물이다. 이런 작제물은 서열 번호: 805의 핵산 서열을 가질 수 있거나, 또는 서열 번호: 805의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다 (하기 참조). 서열 번호: 805 또는 이의 변이체의 발현 작제물은 서열 번호: 806의 핵산 서열 또는 서열 번호: 806의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 벡터 내로 통합될 수 있다.
(서열 번호: 805)
핵심: 단일 밑줄: 프로모터 서열; 굵은 글씨: 가이드 및 승객 서열을 내포하는 마이크로RNA 스템 루프 구조; 이중 밑줄: 폴리A 서열; 이탤릭체: 스터퍼 서열 1 (서열 번호: 815); 이탤릭체+밑줄 : 스터퍼 서열 2 (서열 번호: 816).
대안으로, ASO 서열 MW (서열 번호: 80)를 내포하는 안티센스 작제물은 마이크로RNA 코딩 서열이 서열 번호: 800의 핵산 서열을 갖는 폴리뉴클레오티드이거나, 또는 서열 번호: 800의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이도록, E-miR-124-3 골격을 포함할 수 있다.
(서열 번호: 800)
핵심: 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+물결선 밑줄: MW 승객 서열을 인코딩하는 DNA; 이탤릭체+밑줄+회색 강조 : MW 가이드 서열을 인코딩하는 DNA.
서열 번호: 800의 작제물은 아래의 서열 번호: 821에 나타나 있는 바와 같이, hSyn 프로모터 (서열 번호: 790) 및 폴리A 서열을 더욱 포함할 수 있다.
(서열 번호: 821)
핵심: 단일 밑줄 = 프로모터 서열; 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+물결선 밑줄: GI 승객 서열을 인코딩하는 DNA; 이탤릭체+굵은 글씨+단일 밑줄 : GI 가이드 서열을 인코딩하는 DNA 이중 밑줄: 폴리A 서열.
본원 발명의 다른 단일시스트론성, 항-Grik2 작제물은 hSyn 프로모터 (서열 번호: 790), E-miR-124-3 골격 내로 통합된 ASO MW (서열 번호: 80), 그리고 하나 이상의 스터퍼 서열 (서열 번호: 815 및/또는 서열 번호: 816)을 내포하는 AAV (예를 들면, AAV9) 작제물이다. 이런 작제물은 서열 번호: 807의 핵산 서열을 가질 수 있거나, 또는 서열 번호: 807의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다 (하기 참조). 서열 번호: 807 또는 이의 변이체의 발현 작제물은 서열 번호: 808의 핵산 서열 또는 서열 번호: 808의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 벡터 내로 통합될 수 있다.
(서열 번호: 807)
핵심: 단일 밑줄: 프로모터 서열; 굵은 글씨: 가이드 및 승객 서열을 내포하는 마이크로RNA 스템 루프 구조; 이중 밑줄: 폴리A 서열; 이탤릭체: 스터퍼 서열 1 (서열 번호: 815); 이탤릭체+밑줄 : 스터퍼 서열 2 (서열 번호: 816).
다중유전자 miRNA 카세트
측부/스템 루프/측부 작제물 (예를 들면, 일차-miRNA)은 단일 miRNA "카세트"로서 처리될 수 있고 연쇄체화될 수 있다 (예를 들면, 하나 이상의 프로모터에 의해 주동된 다중유전자 배열로 제공될 수 있다). 하나 초과 (예를 들면, 적어도 2, 3, 4, 5, 6, 7, 8, 9, 10개, 또는 초과)의 프리-miR 스템 루프 서열이 더 긴 전사체 (예컨대 예를 들면, 인트론)의 임의적인 폴리뉴클레오티드 서열 내에 또는 내인성 마이크로RNA 측접 서열 (각 스템 루프의 5'와 3', 예컨대 -5p 및 -3p 서열) 사이에 임베드될 수 있다. 각 프리-miR 스템 루프 서열은 전용 프로모터의 제어하에 발현될 수 있다 (예를 들면, 개별 프리-miR 스템 루프 서열의 발현을 각각 독립적으로 조절하는 별개의 프로모터 서열을 갖는 다중유전자 작제물로서; 다시 말하면, 각 프로모터는 개별 마이크로RNA를 생산하기 위해 다른 것들과 독립적으로 기능한다). 적어도 5-bp-확장된 스템을 제공할 수 있는 측접 서열이 스템 루프의 처리에 충분한 것으로 밝혀졌다 (Sun, et al. BioTechniques .41:59-63, July 2006, 본원에서 참조로서 편입됨). 스페이서 서열은 첫 번째 miRNA 발현 카세트의 3' 측부 서열 및 두 번째 miRNA 발현 카세트의 5' 측부 서열 사이에 배치될 수 있다. 스페이서 서열은 코딩 또는 비코딩 (예를 들면, 인트론) 서열로부터 유래될 수 있고 다양한 길이이지만, 스템 루프-측부 서열의 일부로서 고려되지 않는다 (Rousset, F. et al., Molecular Therapy: Nucleic Acids, 14:352-63, 2019, 본원에서 참조로서 편입됨).
예시적인 발현 카세트는 (a) Grik2 mRNA에 혼성화하는 가이드 RNA 서열을 내포하는 첫 번째 miRNA 서열을 인코딩하는 첫 번째 폴리뉴클레오티드; 및 (b) Grik2 mRNA에 혼성화하는 가이드 RNA 서열을 내포하는 두 번째 miRNA 서열을 인코딩하는 두 번째 폴리뉴클레오티드를 내포하는 뉴클레오티드 서열을 포함할 수 있다. 예를 들면, 발현 카세트는 5'에서 3'로, (a) 가이드 가닥의 5'에 위치된 첫 번째 5' 측부 영역으로서, 상기 첫 번째 측부 영역은 첫 번째 5' 측부 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 및 768 중에서 하나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)을 포함하고; (b) (i) 표 2 및/또는 표 3에서 열거된 가이드 서열 (예를 들면, 서열 번호: 1-100) 중에서 하나에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 가이드 뉴클레오티드 서열을 포함하는 5' 스템 루프 팔; (ii) 표 6에서 선택되는 마이크로RNA 서열 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 포함하는 루프 영역; (iii) 가이드 가닥에 상보적이거나 또는 실질적으로 상보적인 승객 뉴클레오티드 서열을 포함하는 3' 스템 루프 팔을 포함하는 첫 번째 스템 루프 구조; (c) 상기 승객 가닥 및 3' 스페이서 서열의 3'에 위치된 첫 번째 3' 측부 영역 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 및 769 중에서 하나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); (d) 가이드 가닥의 5'에 위치된 두 번째 5' 측부 영역 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 및 768 중에서 하나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); (e) (i) 표 2 및/또는 표 3에서 열거된 가이드 서열 (예를 들면, 서열 번호: 1-100) 중에서 하나에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 가이드 뉴클레오티드 서열을 포함하는 5' 스템 루프 팔; (ii) 표 6에서 선택되는 마이크로RNA 서열 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 내포하는 루프 영역; (iii) 가이드 가닥에 상보적이거나 또는 실질적으로 상보적인 승객 뉴클레오티드 서열을 포함하는 3' 스템 루프 팔을 포함하는 두 번째 스템 루프 구조; (f) 승객 가닥의 3'에 위치된 3' 측부 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 및 769 중에서 하나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)을 포함하는 두 번째 3' 측부 영역을 포함할 수 있다.
첫 번째 5' 측부 서열, 첫 번째 3' 측부 서열, 두 번째 5' 측부 서열, 그리고 두 번째 3' 측부 서열은 표 6에서 선택될 수 있다.
이중-miRNA, 단일 프로모터 발현 카세트
다중유전자 또는 다중유전자 rAAV 발현 작제물은 순차적 (예를 들면, 연속한 또는 비연속한) miRNA-인코딩 폴리뉴클레오티드 X1, 예컨대 (X1)n으로 구성되는 도입유전자를 포함할 수 있다. X1 폴리뉴클레오티드는 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나, 가이드 서열에 완전히 또는 실제적으로 상보적인 승객 서열, 표 6에서 열거된 5'와 3' 측부 서열 중에서 하나, 그리고 표 6에서 열거된 루프 서열 중에서 하나를 포함한다. 화학식, (X1)n을 갖는 다중유전자 도입유전자는 프로모터 및 도입유전자가 화학식, 프로모터-(X1)n (여기서 n은 1-10의 정수 (예를 들면, 1, 2, 3, 4, 5, 6, 7, 8 , 9, 또는 10)이다)을 갖도록, 도입유전자의 5' 단부에 배치된 단일 프로모터의 제어하에 있다.
본원 발명의 멀티-마이크로RNA 작제물의 무제한적 실례는 단일 프로모터, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 790), 내인성 (E)-miR-30 골격에 임베드된 GI 안티센스 서열 (서열 번호: 77), 그리고 E-miR-218-1 골격에 임베드된 MW 안티센스 서열 (서열 번호: 80)을 포함한다. 이런 작제물은 서열 번호: 811의 핵산 서열을 가질 수 있거나, 또는 서열 번호: 811의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다 (하기 참조). 서열 번호: 811 또는 이의 변이체의 작제물은 서열 번호: 812의 핵산 서열 또는 서열 번호: 812의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 벡터 내로 통합될 수 있다.
(서열 번호: 811)
핵심: 단일 밑줄: 프로모터 서열; 굵은 글씨: 가이드 및 승객 서열을 내포하는 마이크로RNA 스템 루프 구조; 이중 밑줄: 폴리A 서열; 이탤릭체: 스터퍼 서열 (서열 번호: 815).
이중-miRNA, 이중 프로모터 발현 카세트
하나 초과 (예를 들면, 적어도 2, 3, 4, 5, 6, 7, 8, 9, 10개, 또는 그 초과)의 프리-miR 스템 루프 서열을 내포하는 다중유전자 발현 카세트는 각 개별 프리-miR 스템 루프 서열이 전용 프로모터 서열에 작동가능하게 연결되도록, 각 개별 프리-miR 스템 루프 서열의 발현을 조절하기 위한 하나 초과의 프로모터 서열을 포함할 수 있다. 이런 사례에서, 발현 작제물은 화학식, (프로모터-X1)n의 구조를 특징으로 하는데, 여기서 X1은 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나를 내포하는 폴리뉴클레오티드이고, 그리고 n은 1-10의 정수 (예를 들면, 1, 2, 3, 4, 5, 6, 7, 8, 9, 또는 10)이다. 추가의 조절 요소, 예컨대 인핸서 서열, 종결자 서열, 폴리아데닐화 신호, 인트론 및/또는 이차 구조를 형성할 수 있는 서열, 예컨대 본원에서 개시된 조절 요소 중에서 하나는 프로모터-X1 구조의 5' 단부 및/또는 3' 단부에 작동가능하게 연결될 수 있다.
특정 실례에서, 이중-miRNA 발현 카세트는 각각, 개별 프로모터 서열 (예를 들면, 본원에서 개시된 프로모터 서열)의 제어하에 2개의 프리-miR 스템 루프 서열을 포함한다. 이중-miRNA 카세트에서 2개의 프로모터는 동일한 프로모터 또는 상이한 프로모터일 수 있다.
특정한 실례에서, 이중-miRNA 발현 카세트는 5'에서 3'로, (a) 첫 번째 프로모터 서열 (예를 들면, 본원에서, 예를 들면, 표 5에서 개시된 프로모터 서열 중에서 하나, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나), 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); (b) 첫 번째 승객 뉴클레오티드 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나)의 5'에 위치된 첫 번째 5' 측부 영역; (c) 첫 번째 스템 루프 서열로서, 5'에서 3'로, (i) 첫 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 첫 번째 승객 뉴클레오티드 서열을 포함하는 첫 번째 5' 스템 루프 팔; (ii) 첫 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 또는 770 중에서 하나)을 내포하는 첫 번째 루프 영역; (iii) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 첫 번째 가이드 뉴클레오티드 서열을 내포하는 첫 번째 3' 스템 루프 팔을 포함하는 첫 번째 스템 루프 서열; (d) 첫 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 또는 769 중에서 하나)의 3'에 위치된 첫 번째 3' 측부 영역; (e) 임의적으로, 두 번째 프로모터 서열 (예를 들면, 본원에서, 예를 들면, 표 5에서 개시된 프로모터 서열 중에서 하나, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나), 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); (f) 두 번째 승객 뉴클레오티드 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나)의 5'에 위치된 두 번째 5' 측부 영역; (g) 두 번째 스템 루프 서열로서, 5'에서 3'로, (i) 두 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 두 번째 승객 뉴클레오티드 서열을 내포하는 두 번째 5' 스템 루프 팔; (ii) 두 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 또는 770 중에서 하나)을 내포하는 두 번째 루프 영역; (iii) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 두 번째 가이드 뉴클레오티드 서열을 내포하는 두 번째 3' 스템 루프 팔을 포함하는 두 번째 스템 루프 서열; 그리고 (h) 두 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 또는 769 중에서 하나)의 3'에 위치된 두 번째 3' 측부 영역을 포함하는 뉴클레오티드 서열을 포함한다.
다른 실례에서, 이중-miRNA 발현 카세트는 5'에서 3'로, (a) 첫 번째 프로모터 서열 (예를 들면, 본원에서, 예를 들면, 표 5에서 개시된 프로모터 서열 중에서 하나, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나), 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); (b) 첫 번째 승객 뉴클레오티드 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나)의 5'에 위치된 첫 번째 5' 측부 영역; (c) 첫 번째 스템 루프 서열로서, 5'에서 3'로, (i) 첫 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 첫 번째 승객 뉴클레오티드 서열을 내포하는 첫 번째 5' 스템 루프 팔; (ii) 첫 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 또는 770 중에서 하나)을 내포하는 첫 번째 루프 영역; (iii) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 첫 번째 가이드 뉴클레오티드 서열을 내포하는 첫 번째 3' 스템 루프 팔을 포함하는 첫 번째 스템 루프 서열; (d) 첫 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 또는 769 중에서 하나)의 3'에 위치된 첫 번째 3' 측부 영역; (e) 임의적으로, 두 번째 프로모터 서열 (예를 들면, 본원에서, 예를 들면, 표 5에서 개시된 프로모터 서열 중에서 하나, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나), 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); (f) 두 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나)의 5'에 위치된 두 번째 5' 측부 영역; (g) 두 번째 스템 루프 서열로서, 5'에서 3'로, (i) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 가이드 뉴클레오티드 서열을 내포하는 두 번째 5' 스템 루프 팔; (ii) 두 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 또는 770 중에서 하나)을 내포하는 두 번째 루프 영역; (iii) 두 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 두 번째 승객 뉴클레오티드 서열을 내포하는 두 번째 3' 스템 루프 팔을 포함하는 두 번째 스템 루프 서열; 그리고 (h) 두 번째 승객 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 또는 769 중에서 하나)의 3'에 위치된 두 번째 3' 측부 영역을 포함하는 뉴클레오티드 서열을 포함한다.
다른 실례에서, 이중-miRNA 발현 카세트는 5'에서 3'로, (a) 첫 번째 프로모터 서열 (예를 들면, 본원에서, 예를 들면, 표 5에서 개시된 프로모터 서열 중에서 하나, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나), 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); (b) 첫 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나)의 5'에 위치된 첫 번째 5' 측부 영역; (c) 첫 번째 스템 루프 서열로서, 5'에서 3'로, (i) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 첫 번째 가이드 뉴클레오티드 서열을 내포하는 첫 번째 5' 스템 루프 팔; (ii) 첫 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 또는 770 중에서 하나)을 내포하는 첫 번째 루프 영역; (iii) 첫 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 첫 번째 승객 뉴클레오티드 서열을 내포하는 첫 번째 3' 스템 루프 팔을 포함하는 첫 번째 스템 루프 서열; (d) 첫 번째 승객 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 또는 769 중에서 하나)의 3'에 위치된 첫 번째 3' 측부 영역; (e) 임의적으로, 두 번째 프로모터 서열 (예를 들면, 본원에서, 예를 들면, 표 5에서 개시된 프로모터 서열 중에서 하나, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나), 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); (f) 두 번째 승객 뉴클레오티드 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나)의 5'에 위치된 두 번째 5' 측부 영역; (g) 두 번째 스템 루프 서열로서, 5'에서 3'로, (i) 두 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 두 번째 승객 뉴클레오티드 서열을 내포하는 두 번째 5' 스템 루프 팔; (ii) 두 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 또는 770 중에서 하나)을 내포하는 두 번째 루프 영역; (iii) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 두 번째 가이드 뉴클레오티드 서열을 내포하는 두 번째 3' 스템 루프 팔을 포함하는 두 번째 스템 루프 서열; 그리고 (h) 두 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 또는 769 중에서 하나)의 3'에 위치된 두 번째 3' 측부 영역을 포함하는 뉴클레오티드 서열을 포함한다.
다른 실례에서, 이중-miRNA 발현 카세트는 5'에서 3'로, (a) 첫 번째 프로모터 서열 (예를 들면, 본원에서, 예를 들면, 표 5에서 개시된 프로모터 서열 중에서 하나, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나), 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); (b) 첫 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나)의 5'에 위치된 첫 번째 5' 측부 영역; (c) 첫 번째 스템 루프 서열로서, 5'에서 3'로, (i) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 첫 번째 가이드 뉴클레오티드 서열을 내포하는 첫 번째 5' 스템 루프 팔; (ii) 첫 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 또는 770 중에서 하나)을 내포하는 첫 번째 루프 영역; (iii) 첫 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 첫 번째 승객 뉴클레오티드 서열을 내포하는 첫 번째 3' 스템 루프 팔을 포함하는 첫 번째 스템 루프 서열; (d) 첫 번째 승객 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 또는 769 중에서 하나)의 3'에 위치된 첫 번째 3' 측부 영역; (e) 임의적으로, 두 번째 프로모터 서열 (예를 들면, 본원에서, 예를 들면, 표 5에서 개시된 프로모터 서열 중에서 하나, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나), 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); (f) 두 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나)의 5'에 위치된 두 번째 5' 측부 영역; (g) 두 번째 스템 루프 서열로서, 5'에서 3'로, (i) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 가이드 뉴클레오티드 서열을 내포하는 두 번째 5' 스템 루프 팔; (ii) 두 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 또는 770 중에서 하나)을 내포하는 두 번째 루프 영역; (iii) 두 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 두 번째 승객 뉴클레오티드 서열을 내포하는 두 번째 3' 스템 루프 팔을 포함하는 두 번째 스템 루프 서열; 그리고 (h) 두 번째 승객 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 또는 769 중에서 하나)의 3'에 위치된 두 번째 3' 측부 영역을 포함하는 뉴클레오티드 서열을 포함한다.
다른 실례에서, 이중-miRNA 발현 카세트는 5'에서 3'로, (a) 5' ITR 서열 (예를 들면, 서열 번호: 746 또는 서열 번호: 747의 핵산 서열, 또는 서열 번호: 746 또는 서열 번호: 747의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 폴리뉴클레오티드); (b) 첫 번째 프로모터 서열 (예를 들면, 본원의 표 5에서 개시된 프로모터 서열 중에서 하나, 또는 표 5에서 열거된 프로모터 서열, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나) 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); (c) 첫 번째 승객 뉴클레오티드 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나, 또는 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)의 5'에 위치된 첫 번째 5' 측부 영역; (d) 첫 번째 스템 루프 서열로서, 5'에서 3'로, (i) 첫 번째 가이드 서열에 상보적이거나 또는 실제적으로 상보적인 첫 번째 승객 뉴클레오티드 서열을 내포하는 첫 번째 5' 스템 루프 팔; (ii) 첫 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 또는 770 중에서 하나, 또는 서열 번호: 758, 761, 764, 767 또는 770에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)을 내포하는 첫 번째 루프 영역; (iii) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 첫 번째 가이드 뉴클레오티드 서열을 내포하는 첫 번째 3' 스템 루프 팔을 포함하는 첫 번째 스템 루프 서열; (e) 첫 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 또는 769 중에서 하나, 또는 서열 번호: 753, 755, 757, 760, 763, 766 또는 769에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)의 3'에 위치된 첫 번째 3' 측부 영역; (f) BGH 폴리A 서열 (예를 들면, 서열 번호: 793 또는 서열 번호: 793에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체), (g) 임의적으로, 두 번째 프로모터 서열 (예를 들면, 본원의 표 5에서 개시된 프로모터 서열 중에서 하나, 또는 표 5에서 열거된 프로모터 서열, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나) 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); (h) 두 번째 승객 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나, 또는 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)의 5'에 위치된 두 번째 5' 측부 영역; (i) 두 번째 스템 루프 서열로서, 5'에서 3'로, (i) 두 번째 가이드 서열에 상보적이거나 또는 실제적으로 상보적인 두 번째 승객 뉴클레오티드 서열을 내포하는 두 번째 5' 스템 루프 팔; (ii) 두 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 또는 770 중에서 하나, 또는 서열 번호: 758, 761, 764, 767 또는 770에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)을 내포하는 두 번째 루프 영역; (iii) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 가이드 뉴클레오티드 서열을 내포하는 두 번째 3' 스템 루프 팔을 포함하는 두 번째 스템 루프 서열; 및 (j) 두 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 또는 769 중에서 하나, 또는 서열 번호: 753, 755, 757, 760, 763, 766 또는 769에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)의 3'에 위치된 두 번째 3' 측부 영역; (k) RBG 폴리A 서열 (예를 들면, 서열 번호: 750, 서열 번호: 751 또는 서열 번호: 792, 또는 서열 번호: 750, 서열 번호: 751 또는 서열 번호: 792에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); 그리고 3' ITR 서열 (예를 들면, 서열 번호: 748, 서열 번호: 749 또는 서열 번호: 789, 또는 서열 번호: 748, 서열 번호: 749 또는 서열 번호: 789에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)을 포함하는 뉴클레오티드 서열을 포함한다.
다른 실례에서, 이중-miRNA 발현 카세트는 5'에서 3'로, (a) 5' ITR 서열 (예를 들면, 서열 번호: 746 또는 서열 번호: 747의 핵산 서열, 또는 서열 번호: 746 또는 서열 번호: 747의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 폴리뉴클레오티드); (b) 첫 번째 프로모터 서열 (예를 들면, 본원의 표 5에서 개시된 프로모터 서열 중에서 하나, 또는 표 5에서 열거된 프로모터 서열, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나) 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); (c) 첫 번째 승객 뉴클레오티드 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나, 또는 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)의 5'에 위치된 첫 번째 5' 측부 영역; (d) 첫 번째 스템 루프 서열로서, 5'에서 3'로, (i) 첫 번째 가이드 서열에 상보적이거나 또는 실제적으로 상보적인 첫 번째 승객 뉴클레오티드 서열을 내포하는 첫 번째 5' 스템 루프 팔; (ii) 첫 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 또는 770 중에서 하나, 또는 서열 번호: 758, 761, 764, 767 또는 770에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)을 내포하는 첫 번째 루프 영역; (iii) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 첫 번째 가이드 뉴클레오티드 서열을 내포하는 첫 번째 3' 스템 루프 팔을 포함하는 첫 번째 스템 루프 서열; (e) 첫 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 또는 769 중에서 하나, 또는 서열 번호: 753, 755, 757, 760, 763, 766 또는 769에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)의 3'에 위치된 첫 번째 3' 측부 영역; (f) BGH 폴리A 서열 (예를 들면, 서열 번호: 793 또는 서열 번호: 793에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체), (g) 임의적으로, 두 번째 프로모터 서열 (예를 들면, 본원의 표 5에서 개시된 프로모터 서열 중에서 하나, 또는 표 5에서 열거된 프로모터 서열, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나) 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); (h) 두 번째 승객 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나, 또는 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)의 5'에 위치된 두 번째 5' 측부 영역; (i) 두 번째 스템 루프 서열로서, 5'에서 3'로, (i) 두 번째 가이드 서열에 상보적이거나 또는 실제적으로 상보적인 두 번째 승객 뉴클레오티드 서열을 내포하는 두 번째 5' 스템 루프 팔; (ii) 두 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 또는 770 중에서 하나, 또는 서열 번호: 758, 761, 764, 767 또는 770에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)을 내포하는 두 번째 루프 영역; (iii) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 가이드 뉴클레오티드 서열을 내포하는 두 번째 3' 스템 루프 팔을 포함하는 두 번째 스템 루프 서열; 및 (j) 두 번째 승객 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 또는 769 중에서 하나, 또는 서열 번호: 753, 755, 757, 760, 763, 766 또는 769에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)의 3'에 위치된 두 번째 3' 측부 영역; (k) RBG 폴리A 서열 (예를 들면, 서열 번호: 750, 서열 번호: 751 또는 서열 번호: 792, 또는 서열 번호: 750, 서열 번호: 751 또는 서열 번호: 792에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); 그리고 3' ITR 서열 (예를 들면, 서열 번호: 748, 서열 번호: 749 또는 서열 번호: 789, 또는 서열 번호: 748, 서열 번호: 749 또는 서열 번호: 789에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)을 포함하는 뉴클레오티드 서열을 포함한다.
다른 실례에서, 이중-miRNA 발현 카세트는 5'에서 3'로, (a) 5' ITR 서열 (예를 들면, 서열 번호: 746 또는 서열 번호: 747의 핵산 서열, 또는 서열 번호: 746 또는 서열 번호: 747의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 폴리뉴클레오티드); (b) 첫 번째 프로모터 서열 (예를 들면, 본원의 표 5에서 개시된 프로모터 서열 중에서 하나, 또는 표 5에서 열거된 프로모터 서열, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나) 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); (c) 첫 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나, 또는 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)의 5'에 위치된 첫 번째 5' 측부 영역; (d) 첫 번째 스템 루프 서열로서, 5'에서 3'로, (i) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 첫 번째 가이드 뉴클레오티드 서열을 내포하는 첫 번째 5' 스템 루프 팔; (ii) 첫 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 또는 770 중에서 하나, 또는 서열 번호: 758, 761, 764, 767 또는 770에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)을 내포하는 첫 번째 루프 영역; (iii) 첫 번째 가이드 서열에 상보적이거나 또는 실제적으로 상보적인 첫 번째 승객 뉴클레오티드 서열을 내포하는 첫 번째 3' 스템 루프 팔을 포함하는 첫 번째 스템 루프 서열; (e) 첫 번째 승객 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 또는 769 중에서 하나, 또는 서열 번호: 753, 755, 757, 760, 763, 766 또는 769에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)의 3'에 위치된 첫 번째 3' 측부 영역; (f) BGH 폴리A 서열 (예를 들면, 서열 번호: 793 또는 서열 번호: 793에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체), (g) 임의적으로, 두 번째 프로모터 서열 (예를 들면, 본원의 표 5에서 개시된 프로모터 서열 중에서 하나, 또는 표 5에서 열거된 프로모터 서열, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나) 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); (h) 두 번째 승객 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나, 또는 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)의 5'에 위치된 두 번째 5' 측부 영역; (i) 두 번째 스템 루프 서열로서, 5'에서 3'로, (i) 두 번째 가이드 서열에 상보적이거나 또는 실제적으로 상보적인 두 번째 승객 뉴클레오티드 서열을 내포하는 두 번째 5' 스템 루프 팔; (ii) 두 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 또는 770 중에서 하나, 또는 서열 번호: 758, 761, 764, 767 또는 770에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)을 내포하는 두 번째 루프 영역; (iii) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 가이드 뉴클레오티드 서열을 내포하는 두 번째 3' 스템 루프 팔을 포함하는 두 번째 스템 루프 서열; 및 (j) 두 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 또는 769 중에서 하나, 또는 서열 번호: 753, 755, 757, 760, 763, 766 또는 769에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)의 3'에 위치된 두 번째 3' 측부 영역; (k) RBG 폴리A 서열 (예를 들면, 서열 번호: 750, 서열 번호: 751 또는 서열 번호: 792, 또는 서열 번호: 750, 서열 번호: 751 또는 서열 번호: 792에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); 그리고 3' ITR 서열 (예를 들면, 서열 번호: 748, 서열 번호: 749 또는 서열 번호: 789, 또는 서열 번호: 748, 서열 번호: 749 또는 서열 번호: 789에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)을 포함하는 뉴클레오티드 서열을 포함한다.
다른 실례에서, 이중-miRNA 발현 카세트는 5'에서 3'로, (a) 5' ITR 서열 (예를 들면, 서열 번호: 746 또는 서열 번호: 747의 핵산 서열, 또는 서열 번호: 746 또는 서열 번호: 747의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 폴리뉴클레오티드); (b) 첫 번째 프로모터 서열 (예를 들면, 본원의 표 5에서 개시된 프로모터 서열 중에서 하나, 또는 표 5에서 열거된 프로모터 서열, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나) 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); (c) 첫 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나, 또는 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)의 5'에 위치된 첫 번째 5' 측부 영역; (d) 첫 번째 스템 루프 서열로서, 5'에서 3'로, (i) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 첫 번째 가이드 뉴클레오티드 서열을 내포하는 첫 번째 5' 스템 루프 팔; (ii) 첫 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 또는 770 중에서 하나, 또는 서열 번호: 758, 761, 764, 767 또는 770에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)을 내포하는 첫 번째 루프 영역; (iii) 첫 번째 가이드 서열에 상보적이거나 또는 실제적으로 상보적인 첫 번째 승객 뉴클레오티드 서열을 내포하는 첫 번째 3' 스템 루프 팔을 포함하는 첫 번째 스템 루프 서열; (e) 첫 번째 승객 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 또는 769 중에서 하나, 또는 서열 번호: 753, 755, 757, 760, 763, 766 또는 769에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)의 3'에 위치된 첫 번째 3' 측부 영역; (f) BGH 폴리A 서열 (예를 들면, 서열 번호: 793 또는 서열 번호: 793에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체), (g) 임의적으로, 두 번째 프로모터 서열 (예를 들면, 본원의 표 5에서 개시된 프로모터 서열 중에서 하나, 또는 표 5에서 열거된 프로모터 서열, 예를 들면, hSyn 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나), CaMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나) 또는 C1ql2 프로모터 (예를 들면, 서열 번호: 719 또는 서열 번호: 791), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); (h) 두 번째 가이드 뉴클레오티드 서열 (예를 들면, 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나, 또는 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)의 5'에 위치된 두 번째 5' 측부 영역; (i) 두 번째 스템 루프 서열로서, 5'에서 3'로, (i) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나 (예를 들면, G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80) 또는 MU (서열 번호: 96), 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 가이드 뉴클레오티드 서열을 내포하는 두 번째 5' 스템 루프 팔; (ii) 두 번째 마이크로RNA 루프 서열 (예를 들면, 서열 번호: 758, 761, 764, 767 또는 770 중에서 하나, 또는 서열 번호: 758, 761, 764, 767 또는 770에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)을 내포하는 두 번째 루프 영역; (iii) 두 번째 가이드 서열에 상보적이거나 또는 실제적으로 상보적인 두 번째 승객 뉴클레오티드 서열을 내포하는 두 번째 3' 스템 루프 팔을 포함하는 두 번째 스템 루프 서열; 및 (j) 두 번째 승객 뉴클레오티드 서열 (예를 들면, 서열 번호: 753, 755, 757, 760, 763, 766 또는 769 중에서 하나, 또는 서열 번호: 753, 755, 757, 760, 763, 766 또는 769에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)의 3'에 위치된 두 번째 3' 측부 영역; (k) RBG 폴리A 서열 (예를 들면, 서열 번호: 750, 서열 번호: 751 또는 서열 번호: 792, 또는 서열 번호: 750, 서열 번호: 751 또는 서열 번호: 792에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체); 그리고 3' ITR 서열 (예를 들면, 서열 번호: 748, 서열 번호: 749 또는 서열 번호: 789, 또는 서열 번호: 748, 서열 번호: 749 또는 서열 번호: 789에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체)을 포함하는 뉴클레오티드 서열을 포함한다.
한 가지 실례에서, 첫 번째 가이드 서열은 G9 서열 (서열 번호: 68)이거나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이고, 그리고 두 번째 가이드 서열은 GI 서열 (서열 번호: 77)이거나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이다. 다른 실례에서, 첫 번째 가이드 서열은 G9 서열 (서열 번호: 68이거나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이고, 그리고 두 번째 가이드 서열은 MW 서열 (서열 번호: 80)이거나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이다. 다른 실례에서, 첫 번째 가이드 서열은 GI 서열 (서열 번호: 77)이거나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이고, 그리고 두 번째 가이드 서열은 G9 서열 (서열 번호: 68)이거나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이다. 또 다른 실례에서, 첫 번째 가이드 서열은 GI 서열 (서열 번호: 77)이거나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이고, 그리고 두 번째 가이드 서열은 MW 서열 (서열 번호: 80)이거나 또는 거기에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이다.
다른 실례에서, 첫 번째 프로모터는 SYN 프로모터 (예를 들면, 서열 번호: 682-685 및 790 중에서 하나 또는 거기에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이고, 그리고 임의적으로, 두 번째 프로모터는 CAMKII 프로모터 (예를 들면, 서열 번호: 687-691 및 802 중에서 하나 또는 거기에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체이다.
전술된 이중-miRNA 발현 카세트에서 설명된 서열에 대한 서열 동일성은 10-1500 (예를 들면, 20-1400, 30-1300, 40-1200, 50-1100, 60-1000, 70-900, 80-800, 90-700, 100-600, 200-500, 또는 300-400)개 뉴클레오티드의 범위에 걸쳐 결정될 수 있다. 예를 들면, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 10개의 뉴클레오티드에 걸쳐 결정될 수 있다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 20개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 30개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 40개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 50개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 60개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 70개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 80개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 90개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 100개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 150개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 200개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 250개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 300개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 350개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 400개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 450개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 500개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 550개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 600개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 650개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 700개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 750개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 800개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 850개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 900개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 950개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 1000개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 1100개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 1200개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 1300개의 뉴클레오티드에 걸쳐 결정된다. 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 1400개의 뉴클레오티드에 걸쳐 결정된다. 또 다른 실례에서, 전술된 이중-miRNA 발현 카세트에 대한 서열 동일성은 1500개의 뉴클레오티드에 걸쳐 결정된다.
전술된 이중-miRNA 발현 카세트는 U6 프로모터, H1 프로모터, 7SK 프로모터, 아포지질단백질 E-인간 알파 1-항트립신 프로모터, CAG 프로모터, CBA 프로모터, CK8 프로모터, mU1a 프로모터, 신장 인자 1α 프로모터, 티록신 결합 글로불린 프로모터, 시냅신 프로모터, RNA 결합 Fox-1 동족체 3 프로모터, 칼슘/칼모듈린 의존성 단백질 키나아제 II 프로모터, 뉴런 특이적 에놀라아제 프로모터, 혈소판 유래 성장 인자 아단위 β, 소포성 글루타민산염 전달체 프로모터, 소마토스타틴 프로모터, 신경펩티드 Y 프로모터, 혈관작용 장관 펩티드 프로모터, 파브알부민 프로모터, 글루타민산염 탈카르복실화효소 65 프로모터, 글루타민산염 탈카르복실화효소 67 프로모터, 도파민 수용체 D1 프로모터, 도파민 수용체 D2 프로모터, 보체 C1q 유사 2 프로모터, 프로오피오멜라노코르틴 프로모터, 프로스페로 호메오복스 1 프로모터, 미소관 관련 단백질 1B 프로모터, 그리고 튜불린 알파 1 프로모터로 구성된 군에서 선택되는 프로모터를 포함할 수 있다.
본원에서 개시된 이중-miRNA 발현 카세트와 함께 이용하는 데 적합한 마이크로RNA 루프 서열은 miR-30, miR-155, miR-218-1, 또는 miR-124-3 루프 서열일 수 있다.
본원 발명의 이중-miRNA 발현 카세트는 또한, 발현 카세트의 5' 단부 상에서 5'-ITR (예를 들면, 서열 번호: 746 또는 서열 번호: 747) 및 발현 카세트의 3' 단부 상에서 3'-ITR (예를 들면, 서열 번호: 748, 749 및 789 중에서 하나)을 통합할 수 있다.
게다가, 본원에서 개시된 이중-miRNA 발현 작제물은 첫 번째 3' 측부 영역의 3' 단부 및 두 번째 프로모터의 5' 단부 사이에 작동가능하게 연결된 첫 번째 폴리아데닐화 (폴리A) 신호 및/또는 두 번째 3' 측부 영역의 두 번째 3' 단부 및 3' ITR 사이에 작동가능하게 연결된 두 번째 폴리A 신호를 포함할 수 있다. 첫 번째 폴리A 신호 및 두 번째 폴리A 신호는 동일할 수 있거나 (예를 들면, 둘 모두 RBG 또는 BGH 폴리A 신호이다) 또는 상이할 수 있다 (예를 들면, 첫 번째 폴리A 신호는 RBG 폴리A이고 두 번째 폴리A 신호는 BGH 폴리A이거나; 또는 첫 번째 폴리A 신호는 BGH 폴리A이고 두 번째 폴리A 신호는 RBG 폴리A이다).
본원 발명의 예시적인 이중-miRNA, 항-Grik2 작제물은 A-miR-30 골격 내로 통합된 G9 (서열 번호: 68) ASO 서열에 작동가능하게 연결된 hSyn 프로모터 (서열 번호: 790), 그 이후에 A-miR-30 골격 내로 통합된 GI (서열 번호: 77) ASO 서열에 작동가능하게 연결된 CaMKII 프로모터 (서열 번호: 802)를 내포하는 AAV (예를 들면, AAV9) 작제물을 포함할 수 있다 (DMTPV1). 이런 작제물은 서열 번호: 785의 핵산 서열을 가질 수 있거나, 또는 서열 번호: 785의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다 (하기 참조).
(서열 번호: 785)
핵심: 굵은 글씨 = 5' ITR 서열; 단일 밑줄 = 프로모터 서열; 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+물결선 밑줄: 가이드 서열 (G9 및 GI - 상기 순서로)에 상보적인 승객 서열을 인코딩하는 DNA; 이탤릭체+굵은 글씨+단일 밑줄 : 가이드 서열 (G9 및 GI - 상기 순서로)을 인코딩하는 DNA; 이중 밑줄: 폴리A 서열; 굵은 글씨+소문자: 3' ITR 서열 (서열 번호: 789).
본원 발명의 다른 예시적인 이중-miRNA, 항-Grik2 작제물은 E-miR-124-3 골격 내로 통합된 G9 (서열 번호: 68) ASO 서열에 작동가능하게 연결된 hSyn 프로모터 (서열 번호: 790), 그 이후에 E-miR-124-3 골격 내로 통합된 MW (서열 번호: 80) ASO 서열에 작동가능하게 연결된 CaMKII 프로모터 (서열 번호: 802)를 내포하는 AAV (예를 들면, AAV9) 작제물을 포함할 수 있다 (DMTPV2). 이런 작제물은 서열 번호: 786의 핵산 서열을 가질 수 있거나, 또는 서열 번호: 786의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다 (하기 참조).
(서열 번호: 786)
핵심: 굵은 글씨 = 5' ITR 서열; 단일 밑줄 = 프로모터 서열; 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+물결선 밑줄: 가이드 서열 (G9 및 MW - 상기 순서로)에 상보적인 승객 서열을 인코딩하는 DNA; 이탤릭체+굵은 글씨+단일 밑줄 : 가이드 서열 (G9 및 MW - 상기 순서로)을 인코딩하는 DNA; 이중 밑줄: 폴리A 서열; 굵은 글씨+소문자: 3' ITR 서열 (서열 번호: 789).
본원 발명의 다른 예시적인 이중-miRNA, 항-Grik2 작제물은 E-miR-30-3 골격 내로 통합된 GI (서열 번호: 77) ASO 서열에 작동가능하게 연결된 hSyn 프로모터 (서열 번호: 790), 그 이후에 E-miR-124-3 골격 내로 통합된 G9 (서열 번호: 68) ASO 서열에 작동가능하게 연결된 CaMKII 프로모터 (서열 번호: 802)를 내포하는 AAV (예를 들면, AAV9) 작제물을 포함할 수 있다 (DMTPV3). 이런 작제물은 서열 번호: 787의 핵산 서열을 가질 수 있거나, 또는 서열 번호: 787의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다 (하기 참조).
(서열 번호: 787)
핵심: 굵은 글씨 = 5' ITR 서열; 단일 밑줄 = 프로모터 서열; 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+물결선 밑줄: 가이드 서열 (GI 및 G9 - 상기 순서로)에 상보적인 승객 서열을 인코딩하는 DNA; 이탤릭체+굵은 글씨+단일 밑줄 : 가이드 서열 (GI 및 G9 - 상기 순서로)을 인코딩하는 DNA; 이중 밑줄: 폴리A 서열; 굵은 글씨+소문자: 3' ITR 서열 (서열 번호: 789).
본원 발명의 다른 예시적인 이중-miRNA, 항-Grik2 작제물은 E-miR-30-3 골격 내로 통합된 GI (서열 번호: 77) ASO 서열에 작동가능하게 연결된 hSyn 프로모터 (서열 번호: 790), 그 이후에 E-miR-124-3 골격 내로 통합된 MW (서열 번호: 80) ASO 서열에 작동가능하게 연결된 CaMKII 프로모터 (서열 번호: 802)를 내포하는 AAV (예를 들면, AAV9) 작제물을 포함할 수 있다 (DMTPV4). 이런 작제물은 서열 번호: 788의 핵산 서열을 가질 수 있거나, 또는 서열 번호: 788의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다 (하기 참조).
(서열 번호: 788)
핵심: 굵은 글씨 = 5' ITR 서열; 단일 밑줄 = 프로모터 서열; 이탤릭체 = 5' 측부 서열-가이드 서열-마이크로RNA 루프 서열-승객 서열-3' 측부 서열; 이탤릭체+물결선 밑줄: 가이드 서열 (GI 및 MW - 상기 순서로)에 상보적인 승객 서열을 인코딩하는 DNA; 이탤릭체+굵은 글씨+단일 밑줄 : 가이드 서열 (GI 및 MW - 상기 순서로)을 인코딩하는 DNA; 이중 밑줄: 폴리A 서열; 굵은 글씨+소문자: 3' ITR 서열 (서열 번호: 789).
본원 발명의 다른 예시적인 이중-miRNA, 항-Grik2 작제물은 E-miR-30-3 골격 내로 통합된 GI (서열 번호: 77) ASO 서열에 작동가능하게 연결된 hSyn 프로모터 (서열 번호: 790), 그 이후에 E-miR-218-1 골격 내로 통합된 MW (서열 번호: 80) ASO 서열에 작동가능하게 연결된 CaMKII 프로모터 (서열 번호: 802)를 내포하는 AAV (예를 들면, AAV9) 작제물을 포함할 수 있다 (DMTPV5). 이런 작제물은 서열 번호: 813의 핵산 서열을 가질 수 있거나, 또는 서열 번호: 813의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체일 수 있다 (하기 참조). 이런 발현 카세트는 서열 번호: 814의 핵산 서열 또는 서열 번호: 814의 핵산 서열에 적어도 85% (적어도 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는 이의 변이체를 갖는 벡터 내로 통합될 수 있다.
(서열 번호: 813)
핵심: 단일 밑줄: 프로모터 서열; 굵은 글씨: 가이드 및 승객 서열을 내포하는 마이크로RNA 스템 루프 구조; 이중 밑줄: 폴리A 서열; 이탤릭체: 스터퍼 서열 (서열 번호: 816).
복수의 miRNA 서열을 내포하는 AAV 벡터의 제조에서 향상
단일 miRNA 발현 카세트 (예를 들면, 본원에서 개시된 발현 카세트)를 인코딩하는 플라스미드를 이용한 AAV 벡터의 제조는 부적합한 AAV 유전체 포장에 의해 잠재적으로 방해를 받을 수 있다. 첫째, 일차-miRNA 서열은 짧고 (<200개 염기), 그리고 프로모터 길이에 따라서, 단일 miRNA의 발현을 제어하는 단일 프로모터를 갖는 도입유전자 카세트의 설계는 AAV의 최대 포장 용량 (~4.8 kb)보다 훨씬 짧은 AAV 유전체를 야기할 수 있다. 이런 이유로, 만약 기대되는 전체 유전체 길이가 <2.4 kb (AAV의 포장 용량의 절반)이면, 단일 캡시드가 하나 초과의 벡터로 부하되는 것이 가능할 수 있다. 이것은, 만약 적합한 길이이면 AAV 캡시드 내로 포장될 수 있는 AAV 유전체 이합체 (또는 삼합체)의 생산을 가능하게 하는 적절한 엔도뉴클레아제 닉킹 없이, 중합효소 번역 초과에 의해 매개될 수 있다. 이것은 차후에, AAV 벡터 입자의 모집단 내로 유의미한 이질성을 도입하는데, 이것은 의약품의 제조와 특징화를 훨씬 어렵게 만든다.
둘째, shRNA- 및 miRNA 기반 도입유전자는 miRNA 헤어핀의 포함으로 인해, 생래적으로 유의미한 이차 구조를 갖는다. AAV 유전체 내에 이들 내부 이차 구조는 AAV 유전체 복제와 포장 동안 "가성" ITR로서 기능하여, 절두 사건 및 완전과 부분 벡터의 혼합물을 내포하는 AAV 벡터 입자의 이질성 모집단을 야기할 수 있는 것으로 밝혀졌다.
우리는 추가의 서열 (예를 들면, 추가의 프리-miRNA 스템 루프 서열, 두 번째 프로모터 서열, 스터퍼 서열 (예를 들면, 서열 번호: 815 및/또는 서열 번호: 816), 자가 상보성 AAV 벡터를 이용, 기타 등등)로, AAV 포장 용량 미만의 크기를 갖는 AAV 유전체의 패딩이 부분적인 (다시 말하면, 절두된) AAV 유전체의 사본의 통합을 방지함으로써 AAV 포장을 실제적으로 향상시킨다는 것을 발견하였다. 이런 이유로, 본원에서 설명된 작제물은 전술된 서열을 AAV 발현 카세트 내로 통합하여 벡터 크기를 최대 AAV 포장 용량에 더 가까운 값으로 증가시킴으로써, AAV 유전체의 부적합한 포장을 방지한다.
예를 들면, 단일 프로모터의 제어하에 1개의 miRNA 또는 2개의 별개의 프로모터로부터 2개의 miRNA (이중 작제물 접근방식)를 발현하는 작제물이 시험관내/탈체/인실리코 평가에 의해 예측된 바와 같이, 생체내에서 성과를 낼 수 없는 사례가 있을 수 있다. 이들 사례에서, AAV 벡터 입자의 동질성, 전장, 단독적으로 포장된 모집단을 생산하는 유전체 길이를 확립하기 위해 하기 전략이 실행될 수 있다.
첫째, 만약 단일 miRNA "가이드"의 발현이 요망되면, 프로모터.miR 카세트 그 자체를 교란하지 않으면서 AAV 벡터의 전체 길이를 증가시키기 위해 스터퍼 서열 (예를 들면, 서열 번호: 815 및/또는 서열 번호: 816)이 추가될 수 있다. 이러한 스터퍼는 도입유전자 카세트의 하류 (폴리A 서열의 3')에 추가될 수 있다 (예를 들면, 도 6f 및 도 6g 참조). 이러한 스터퍼의 길이와 함량은 AAV 포장 균일성을 향상시키는 능력을 유지하면서 변화될 수 있다. 추가적으로, 이러한 스터퍼는 프로모터의 상류 (5')에 배치될 수 있다. 스터퍼 서열은 scAAV 벡터 또는 단일 가닥 (ss)AAV 벡터의 맥락에서 활용될 수 있다. 일부 구체예에서, 본원 발명의 벡터는 하나 이상의 스터퍼 서열 (예를 들면, 1개, 2개, 또는 그 초과의 스터퍼 서열)을 포함한다. 일부 구체예에서, 하나 이상의 스터퍼 서열은 서열 번호: 815의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는다. 일부 구체예에서, 하나 이상의 스터퍼 서열은 서열 번호: 815의 핵산 서열에 적어도 90% (예를 들면, 적어도 91%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는다. 일부 구체예에서, 하나 이상의 스터퍼 서열은 서열 번호: 815의 핵산 서열에 적어도 95% (예를 들면, 적어도 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는다. 일부 구체예에서, 하나 이상의 스터퍼 서열은 서열 번호: 815의 핵산 서열에 적어도 99% 서열 동일성을 갖는다. 일부 구체예에서, 하나 이상의 스터퍼 서열은 서열 번호: 815의 핵산 서열을 갖는다. 일부 구체예에서, 하나 이상의 스터퍼 서열은 서열 번호: 816의 핵산 서열에 적어도 85% (예를 들면, 적어도 86%, 90%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는다. 일부 구체예에서, 하나 이상의 스터퍼 서열은 서열 번호: 816의 핵산 서열에 적어도 90% (예를 들면, 적어도 91%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는다. 일부 구체예에서, 하나 이상의 스터퍼 서열은 서열 번호: 816의 핵산 서열에 적어도 95% (예를 들면, 적어도 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 갖는다. 일부 구체예에서, 하나 이상의 스터퍼 서열은 서열 번호: 816의 핵산 서열에 적어도 99% 서열 동일성을 갖는다. 일부 구체예에서, 하나 이상의 스터퍼 서열은 서열 번호: 816의 핵산 서열을 갖는다.
둘째, 만약 하나 초과의 miRNA가 발현되지만 단일 프로모터 전략이 선택되면, 벡터는 복수의 miRNA 카세트의 연쇄체화에 의해 제조될 수 있다. 동일한 골격을 이용한 복수의 miRNA 카세트 (예를 들면, 5개까지의 miRNA 카세트)의 연쇄체화는 벡터 내에 상동성 서열 사이에 재조합을 야기할 수 있는 반면 (도 7), 비상동성 측접 및 루프 서열을 갖는 상이한 골격을 이용한 miRNA 카세트의 연쇄체화는 이러한 포장 우려를 개선할 수 있다 (도 8f). 만약 추가의 miRNA 카세트의 포함이 적합한 길이의 벡터를 야기하지 못하면, 길이를 증가시켜 포장 효율을 향상시키기 위해 전술된 바와 같이 스터퍼 서열 (예를 들면, 서열 번호: 815 및/또는 서열 번호: 816)이 통합될 수 있다.
약학적 조성물
본원에서 설명된 올리고뉴클레오티드 또는 이들을 인코딩하는 핵산 벡터는 생체내 투여에 적합한 생물학적으로 양립성 형태로 포유류 (예를 들면, 인간) 개체에게 투여를 위한 약학적 조성물로 조제될 수 있다.
본원에서 개시된 조성물은 개체 (예를 들면, 인간)에게 전달을 위한 임의의 적합한 운반제에서 조제될 수 있다. 예를 들면, 이들은 약학적으로 허용되는 현탁액, 분산액, 용액, 또는 유제에서 조제될 수 있다. 적합한 매질은 식염수 및 리포솜 제조물을 포함한다. 더 특이적으로, 약학적으로 허용되는 운반체는 무균 수성 또는 비수성 용액, 현탁액 및 유제를 포함할 수 있다. 재조합 인간 알부민 (rAlbumin Human NF RECOMBUMIN® Prime) 또한, AAV 벡터 (Albumedix, Nottingham UK)에서 안정제로서 이용될 수 있다. 비수성 용매의 실례는 프로필렌 글리콜, 폴리에틸렌 글리콜, 식물성 오일 예컨대 올리브유, 그리고 주사가능 유기 에스테르 예컨대 에틸 올레산염이다. 수성 운반체는 식염수 및 완충된 매질을 비롯하여, 물, 알코올성/수성 용액, 유제 또는 현탁액을 포함한다. 정맥내 투여에 적합한 운반제는 유체와 영양소 보충물, 전해질 보충물 (예를 들면, 링거의 덱스트로스에 기초된 것들) 등을 포함한다.
보존제 및 다른 첨가제 예컨대 예를 들면, 항균제, 항산화제, 킬레이트화제, 비활성 가스 등이 또한 존재할 수 있다.
표적화된 유전자 전달을 위해 콜로이드 분산 시스템이 또한 이용될 수 있다. 콜로이드 분산 시스템은 거대분자 복합체, 나노캡슐, 마이크로스피어, 비드, 그리고 수중유 유제, 미셀, 혼합된 미셀 및 리포솜을 비롯한 지질 기반 시스템을 포함한다.
본원에서 설명된 조성물은 유리 염기의 형태에서, 염, 용매화합물의 형태에서, 그리고 전구약물로서 이용될 수 있다. 모든 형태는 본원에서 설명된 방법 내에 있다. 본원 발명의 방법에 따라서, 설명된 화합물 또는 염, 용매화합물, 또는 이들의 전구약물은 선택된 투여 루트에 따라서 다양한 형태로 환자에게 투여될 수 있다.
따라서, 본원에서 설명된 조성물은 예를 들면, 경구, 비경구, 척수강내, 뇌실내, 뇌실질내, 협측, 설하, 코, 직장, 패치, 펌프, 또는 경피 투여 및 그에 맞게 조제된 약학적 조성물에 의한 투여용으로 조제될 수 있다. 비경구 투여는 정맥내, 복막내, 피하, 근육내, 경상피, 코, 폐내, 척수강내, 뇌실내, 뇌실질내, 직장 및 국소 투여 방식을 포함한다. 비경구 투여는 선택된 기간에 걸쳐 연속 주입에 의할 수 있다.
본원에서 설명된 작용제의 용액은 계면활성제, 예컨대 히드록시프로필셀룰로오스와 적절하게 혼합된 물에서 제조될 수 있다. 분산액이 또한, 글리세롤, 액체 폴리에틸렌 글리콜, DMSO, 그리고 알코올과 함께 또는 이것 없이 이들의 혼합물에서, 그리고 오일에서 제조될 수 있다. 저장과 이용의 일상적인 조건하에, 이들 제조물은 미생물의 성장을 예방하기 위한 보존제를 내포할 수 있다. 적합한 제제의 선택과 제조를 위한 전통적인 절차 및 성분은 예를 들면 Remington's Pharmaceutical Sciences (2012, 22nd ed.)에서 및 2018년에 공개된 The United States Pharmacopeia: The National Formulary (USP 41 NF 36)에서 설명된다. 주사가능 이용에 적합한 약학적 형태는 무균 주사가능 용액 또는 분산액의 즉석 제조를 위한 무균 수성 용액 또는 분산액 및 무균 분말을 포함한다. 모든 사례에서 상기 형태는 무균이어야 하고, 그리고 주사기를 통해 쉽게 투여될 수 있는 정도까지 유체이어야 한다. 국부, 영역 또는 전신 투여가 또한 적합할 수 있다. 본원에서 설명된 조성물은 유리하게는, 예를 들면, 대략 1 cm 간격으로 이격된, 표적 부위에 주사 또는 복수의 주사를 투여함으로써 접촉될 수 있다.
본원에서 설명된 조성물은 본원에서 언급된 바와 같이, 단독으로 또는 약학적으로 허용되는 운반체와 조합으로 동물, 예를 들면, 인간에게 투여될 수 있는데, 이들의 비율은 화합물의 용해도 및 화학적 특성, 선택된 투여 루트, 그리고 표준 약학적 관례에 의해 결정된다.
따라서, 본원 발명은 본원에서 개시된 ASO 작용제 (예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA 또는 shmiRNA)를 내포하는 약학적 조성물에 관계한다. 특히, 본원 발명은 본원 발명의 ASO 작용제를 포함하는 벡터를 포함하는 조성물에 관계한다. 특정 실례에서, 본원 발명은 본원에서 개시된 바와 같이, 프로모터에 작동가능하게 연결된 본원 발명의 ASO를 포함하는 벡터 (예를 들면, 렌티바이러스 또는 AAV 벡터)를 내포하는 약학적 조성물을 제공한다. 약학적 조성물은 (a) 바이러스 캡시드; 및 (b) AAV ITR과 측접된 발현 카세트를 포함하는 인공 폴리뉴클레오티드를 포함하는 AAV 벡터를 포함할 수 있는데, 여기서 상기 발현 카세트는 CNS 세포에서 폴리뉴클레오티드의 발현을 제어하는 하나 이상의 조절 서열에 작동가능하게 연결된, Grik2 mRNA에 결합하고 이의 발현을 저해하는 올리고뉴클레오티드를 인코딩하는 폴리뉴클레오티드를 포함한다.
본원에서 개시된 ASO 작용제는 약학적 조성물을 형성하기 위해, 약학적으로 허용되는 부형제, 그리고 임의적으로, 지속된 방출 매트릭스, 예컨대 예를 들면, 생물분해성 중합체와 조합될 수 있다. "약학적으로" 또는 "약학적으로 허용되는"은 적합하면, 포유동물, 특히 인간에게 투여될 때 불리한, 알레르기성 또는 다른 부반응을 발생시키지 않는 분자 실체 및 조성물을 지칭한다. 약학적으로 허용되는 운반체 또는 부형제는 임의의 유형의 비독성 고체, 반고체 또는 액체 충전제, 희석제, 캡슐화 물질 또는 제제 보조제를 지칭한다. 본원에서 개시된 약학적 조성물은 뇌내 (예를 들면, 뇌실질내 또는 뇌실내), 근육내, 정맥내, 경피, 국부, 경구, 설하, 피하, 또는 직장 투여용으로 조제될 수 있다. 단독으로 또는 다른 치료제와 조합으로, 조성물의 활성 성분 (예를 들면, Grik2를 표적으로 하는 ASO 작용제)은 전통적인 약학적 지지체와의 혼합물로서 단위 투여 형태로, 치료가 필요한 개체에게 투여될 수 있다. 적합한 단위 투여 형태는 경구 루트 형태 예컨대 정제, 겔 캡슐, 분말, 과립 및 경구 현탁액 또는 용액, 설하 및 협측 투여 형태, 에어로졸, 이식물, 피하, 경피, 국소, 복막내, 근육내, 정맥내, 피하, 경피, 척수강내, 뇌내, 정위 및 비내 투여 형태, 그리고 직장 투여 형태를 포함한다. 전형적으로, 약학적 조성물은 주사될 수 있는 제제에 대해 약학적으로 허용되는 운반제를 내포한다. 이들은 특히 등장성, 무균, 식염수 (모노나트륨 또는 인산이나트륨, 나트륨, 칼륨, 칼슘 또는 염화마그네슘 등, 또는 이런 염의 혼합물), 또는 첨가 시에, 살균된 물 또는 생리식염수의 사례에 따라서, 주사가능 용액의 구성을 허용하는 건성, 특히 동결 건조된 조성물일 수 있다. 주사가능 이용에 적합한 약학적 형태는 무균 수성 용액 또는 분산액, 호마유, 낙화생유 또는 수성 프로필렌 글리콜을 포함하는 제제, 그리고 무균 주사가능 용액 또는 분산액의 즉석 제조를 위한 무균 분말을 포함한다. 모든 사례에서, 상기 형태는 무균이어야 하고, 그리고 쉬운 주사가능성이 가능할 정도까지 유체이어야 한다. 이것은 제조와 저장의 조건하에 안정되어야 하고, 그리고 미생물, 예컨대 세균 및 균류의 오염 작용에 대항하여 보존되어야 한다. 유리 염기 또는 약리학적으로 허용가능 염으로서 본원 발명의 화합물을 포함하는 용액은 계면활성제, 예컨대 히드록시프로필 셀룰로오스와 적절하게 혼합된 물에서 제조될 수 있다. 분산액이 또한, 글리세롤, 액체 폴리에틸렌 글리콜 및 이들의 혼합물에서, 그리고 오일에서 제조될 수 있다. 저장과 이용의 일상적인 조건하에, 이들 제조물은 미생물의 성장을 예방하기 위한 보존제를 내포할 수 있다. 본원에서 개시된 올리고뉴클레오티드 작용제는 중성 또는 염 형태에서 조성물로 조제될 수 있다. 약학적으로 허용되는 염은 산 부가염 (단백질의 자유 아미노 기로 형성됨), 그리고 무기 산 예컨대 예를 들면, 염화수소산 또는 인산, 또는 유기 산 예컨대 아세트산, 옥살산, 주석산, 만델산 등으로 형성된 것들을 포함한다. 자유 카르복실 기로 형성된 염은 또한, 무기 염기 예컨대 예를 들면, 나트륨, 칼륨, 암모늄, 칼슘 또는 수산화제2철, 그리고 유기 염기 예컨대 이소프로필아민, 트리메틸아민, 히스티딘, 프로카인 등으로부터 유래될 수 있다. 운반체는 또한, 예를 들면, 물, 에탄올, 폴리올 (예를 들면, 글리세롤, 프로필렌 글리콜 및 액체 폴리에틸렌 글리콜 등), 이들의 적합한 혼합물, 그리고 식물성 오일을 내포하는 용매 또는 분산 매질일 수 있다. 적절한 유동성은 코팅 (예를 들면, 레시틴)의 이용에 의해, 분산의 경우에 필요한 입자 크기의 유지에 의해, 그리고 계면활성제의 이용에 의해 유지될 수 있다. 미생물의 작용의 예방은 다양한 항균제와 항진균제, 예컨대 예를 들면, 파라벤, 클로로부탄올, 페놀, 소르브산, 티메로살 등에 의해 담보될 수 있다. 일부 경우에, 본원 발명의 약학적 조성물은 등장화제, 예컨대 예를 들면, 당 또는 염화나트륨을 포함할 수 있다. 주사가능 조성물의 연장된 흡수는 흡수를 지연시키는 작용제, 예컨대 예를 들면, 알루미늄 모노스테아레이트 염 및 젤라틴의 이용에 의해 달성될 수 있다. 무균 주사가능 용액은 필요한 양의 활성제를 적합한 용매에서 필요에 따라, 상기 열거된 여러 다른 성분과 통합하고, 그 이후에 여과 살균함으로써 제조된다. 일반적으로, 분산액은 기초 분산 매질 및 본원에서 개시된 것들로부터 필요한 추가 성분을 내포하는 무균 운반제 내로 다양한 살균된 활성 성분을 통합함으로써 제조된다. 무균 주사가능 용액의 제조를 위한 무균 분말의 경우에, 바람직한 제조 방법은 진공 건조와 동결 건조 기술일 수 있는데, 이들은 활성 성분 플러스 이들의 이전에 무균 여과된 용액으로부터 임의의 추가적인 원하는 성분의 분말을 생성한다. 조제 시에, 용액은 투약 요건과 양립하는 방식으로 및 치료적으로 효과적인 양으로 투여될 것이다. 제제는 다양한 약형, 예컨대 전술된 주사가능 용액의 유형으로 쉽게 투여되지만, 약물 방출 캡슐 등이 또한 이용될 수 있다. 수성 용액에서 비경구 투여의 경우에, 예를 들면, 용액은 필요하면 적절하게 완충되어야 하고, 그리고 액체 희석제가 먼저 충분한 식염수 또는 글루코오스와 등장성이 되도록 만들어져야 한다. 이들 특정 수성 용액은 정맥내, 근육내, 피하 및 복막내 투여에 특히 적합하다. 이용될 수 있는 무균 수성 매질은 당해 분야에서 널리 알려져 있다. 예를 들면, 1회 복용량은 1 mL의 등장성 NaCl 용액에서 용해되고, 그리고 1000 mL의 피하 주입 유체에 첨가되거나 또는 주입의 제안된 부위에 주사될 수 있었다. 치료되는 개체의 상태에 따라서 복용량에서 일부 변이가 반드시 발생할 것이다. 어떤 상황에서든, 투여에 대한 책임이 있는 의사는 적합한 환자 정보 및 당해 분야에서 인정된 방법을 이용하여, 개별 개체에 대한 적합한 용량을 결정할 것이다.
진단의 방법
개체 (예를 들면, 인간 개체)는 예를 들면, 당해 분야에서 널리 알려진 방법을 이용하여, 뇌전증 (예를 들면, TLE)을 겪는 것으로 진단될 수 있고, 따라서 본원에서 개시된 조성물과 방법을 이용한 치료가 필요한 것으로 확인될 수 있다. 예를 들면, 개체에서 뇌전증의 진단은 개체의 뇌에서 뇌전증유발 병소 및 뇌전증모양 활성의 심각도를 확인하기 위한 신경생리학적 검사에 의해 인도될 수 있다. 당해 분야에서 널리 알려진 예시적인 신경생리학적 검사 방법은 뇌파검사법 (EEG), 자기뇌파검사 (MEG), 기능적 MRI (fMRI), 단일-광양자 방출 전산화 단층촬영술 (SPECT), 그리고 양전자 방출 단층촬영술 (PET)을 포함한다. EEG 및 MEG는 높은 시간 해상도에서 피질 기능의 연속 척도를 제공하고 발작사이 기간 뇌전증모양 방전의 검출을 가능하게 하는데, 이것은 개체에서 뇌전증 상태의 양성 진단을 표시할 수 있다. 개체에서 뇌전증에 대한 진단을 확정하기 위해, 개체의 연령, 병력 및 생활양식에 적합한 규범 (예를 들면, 참조 모집단, 예컨대 예를 들면, 비-뇌전증 환자 모집단)에 비하여 개체에서 뇌 활동의 비교가 수행될 수 있다.
개체는 TLE (예를 들면, mTLE 또는 lTLE), 양성 롤란드 뇌전증, 전두엽 뇌전증, 영아 연축, 청소년기 근간대 뇌전증, 청소년기 소발작 뇌전증, 아동기 소발작 뇌전증 (피크노렙시), 온수 뇌전증, 레녹스 가스타우트 증후군, 랜도 크레프너 증후군, 드라베 증후군, 진행성 근간대 뇌전증, 반사 뇌전증, 라스무센 증후군, 변연 뇌전증, 뇌전증 지속증, 복부 뇌전증, 거대 양측성 근간대경련, 월경 뇌전증, 작소니언 발작 장애, 라포라 증후군, 그리고 광민감 뇌전증을 포함하지만 이들에 한정되지 않는 다수의 뇌전증 질환 중에서 한 가지를 겪는 것으로 진단될 수 있다. 뇌전증 질환이 TLE인 경우에, TLE는 초점성 또는 전신 발작에 의해 특징화될 수 있다.
개시된 방법을 이용하여, 뇌전증유발 병소를 특정 뇌 영역 (예를 들면, 내측 측두엽, 편측 전두엽, 전두엽 등)으로 국지화하는 것에 근거하여, 환자가 진단될 수 있는 뇌전증의 유형. 뇌전증 뇌 활동의 전기생리학적 시그너처 또한, 개체에서 뇌전증의 특정 유형 또는 아형을 확인하는 데 이용될 수 있다. 예를 들면, 피질 영역 (예를 들면, 해마 또는 대뇌 피질)에서 빠른 (250-600 Hz) 예파 연흔 (SPW-Rs)의 존재는 개체에서 TLE의 양성 진단을 표시할 수 있다. 다른 실례에서, 레녹스 가스타우트 증후군은 종종, 신피질 및 시상의 전역에서 기록된 빠른-실행 전자사진술 진동 (10-15 Hz)의 존재에 의해 특징화된다. 게다가, 만약 환자가 뇌전증 발작의 행동 현성, 예컨대 예를 들면, 대발작, 일시적인 소발작 (~10 초간 지속되는 기간 동안 감소된 의식 수준), 긴장, 근간대경련, 장 또는 방광 제어의 상실, 혀의 깨물기, 피로, 두통, 말하는 데 어려움, 이상 행동 (예를 들면, 움직임이 없는 응시, 또는 손 또는 입의 자동 운동), 정신병 및/또는 국지화된 허약을 공공연히 나타내는 것으로 보이면, 입원환자 시설에서 개체의 비디오 모니터링이 환자에서 뇌전증의 진단을 표시할 수 있다. 뇌전증에 대해 진단되는 개체로부터 자가 기록 증상 또한, 양성 진단을 표시할 수 있다. 이런 자가 기록 증상은 기시감 또는 미시감의 감각, 조짐, 기억상실, 자연발생적이고 이유 없는 공포와 불안, 메스꺼움, 청각, 시각, 후각, 미각 또는 촉각 환각, 시각 왜곡 (예를 들면, 대시증 또는 소시증), 분열 또는 비현실감, 동반감각, 불쾌감 또는 쾌감, 공포, 분노, 또는 형언할 수 없는 감각을 포함할 수 있다.
약학적 용도
전술된 조성물 (예를 들면, ASO 작용제 또는 이것을 인코딩하는 핵산 벡터)의 투여에 의해, 뇌전증 (예를 들면, TLE)으로 진단되거나 또는 뇌전증이 발달할 위험에 처해 있는 개체에서 뇌전증의 치료를 위한 방법이 본원에서 개시된다. 투여 시에, 본원 발명의 ASO 작용제는 Grik2 mRNA에 결합하고 이것의 발현을 저해할 수 있다. 본원에서 개시된 ASO 작용제에 의한 Grik2의 표적화는 첫 번째 세포 또는 세포의 군 (예를 들면, 뉴런 세포; 이런 세포는 예를 들면, 개체 내에 또는 개체로부터 유래된 샘플 내에 존재할 수 있다)에 의해 발현된 Grik2 mRNA의 수준에서 감소에 의해 현성될 수 있는데, 상기 세포는 Grik2가 전사되고 처리되었다 (예를 들면, 상기 세포 또는 세포들을 본원 발명의 올리고뉴클레오티드와 접촉시킴으로써, 또는 본원 발명의 올리고뉴클레오티드를 상기 세포가 존재하거나 또는 존재했던 개체에게 투여함으로써). 특정 실례에서, Grik2의 발현은 첫 번째 세포 또는 세포의 군과 실제적으로 동일하지만 이렇게 처리되지 않은 두 번째 세포 또는 세포의 군 (올리고뉴클레오티드로 처리되지 않은 또는 관심되는 유전자에 표적화된 올리고뉴클레오티드로 처리되지 않은 대조 세포(들))과 비교하여, 첫 번째 세포 또는 세포의 군에서 감소된다. 관심되는 유전자 (예를 들면, Grik2)의 mRNA의 수준에서 감소의 정도는 아래의 면에서 표현될 수 있다:
유전자 (예를 들면, Grik2 유전자)의 발현 수준에서 변화는 관심되는 유전자의 발현에 기능적으로 연결된 파라미터, 예를 들면, 관심되는 유전자의 단백질 발현 또는 단백질의 하류에 신호전달의 감소의 면에서 사정될 수 있다. 관심되는 유전자의 발현 수준에서 변화는 발현 작제물로부터 관심되는 유전자 (내인성 또는 이종성 중 어느 하나)를 발현하는 임의의 세포에서, 그리고 당해 분야에서 알려진 임의의 검정에 의해 결정될 수 있다.
Grik2의 발현 수준에서 변화는 세포 또는 세포의 군에 의해 발현되는 GluK2 단백질의 수준 (예를 들면, 개체로부터 유래된 샘플에서 발현되는 GluK2 단백질의 수준)에서 감소에 의해 현성될 수 있다. Grik2 mRNA 억제의 사정에 대해 앞서 설명된 바와 같이, 처리된 세포 또는 세포의 군에서 GluK2 단백질 발현의 수준에서 변화는 대조 세포 또는 세포의 군에서 단백질의 수준의 백분율로서 유사하게 표현될 수 있다.
Grik2 유전자의 발현에서 변화를 사정하는 데 이용될 수 있는 대조 세포 또는 세포의 군은 본원 발명의 올리고뉴클레오티드와 아직 접촉되지 않은 세포 또는 세포의 군을 포함한다. 예를 들면, 대조 세포 또는 세포의 군은 올리고뉴클레오티드로 치료에 앞서, 개별 개체 (예를 들면, 인간 또는 동물 개체)로부터 유래될 수 있다.
세포 또는 세포의 군에 의해 발현된 Grik2 mRNA의 수준은 mRNA 발현을 사정하기 위한 당해 분야에서 공지된 임의의 방법을 이용하여 결정될 수 있다. 예를 들면, 샘플 내에 Grik2 mRNA의 발현 수준은 전사된 폴리뉴클레오티드, 또는 이의 일부, 예를 들면, mRNA를 검출함으로써 결정될 수 있다. RNA는 예를 들면, 산 페놀/구아니딘 이소티오시안산염 추출 (RNAzol B; Biogenesis), RNEASYTM RNA 제조 키트 (Qiagen) 또는 PAXgene (PreAnalytix, Switzerland)을 이용하는 것을 비롯한, RNA 추출법을 이용하여 세포로부터 추출될 수 있다. 리보핵산 혼성화를 활용하는 전형적인 검정 형식은 핵 런 온 검정, RT-PCR, RNA분해효소 보호 검정, 노던 블로팅, 제자리 혼성화, 그리고 마이크로어레이 분석을 포함한다. 순환성 mRNA는 PCT 공개 WO2012/177906에서 설명된 방법을 이용하여 검출될 수 있는데, 이것의 전체 내용은 본원에서 참조로서 편입된다. 관심되는 유전자의 발현 수준은 또한, 핵산 프로브를 이용하여 결정될 수 있다. 본원에서 이용된 바와 같이, 용어 "프로브"는 특정한 서열, 예를 들면 mRNA에 선택적으로 결합할 수 있는 임의의 분자를 지칭한다. 프로브는 당해 분야의 널리 알려진 전통적인 방법을 이용하여 합성되거나 또는 적합한 생물학적 제조물로부터 유래될 수 있다. 프로브는 표지화되도록 특이적으로 설계될 수 있다. 프로브로서 활용될 수 있는 분자의 실례는 RNA, DNA, 단백질, 항체, 그리고 유기 분자를 포함하지만 이들에 한정되지 않는다.
단리된 mRNA가 서던 또는 노던 분석, 중합효소 연쇄 반응 (PCR) 분석, 그리고 프로브 어레이를 포함하지만 이들에 한정되지 않는 혼성화 또는 증폭 검정에서 이용될 수 있다. mRNA 수준의 결정을 위한 한 가지 방법은 단리된 mRNA를, 관심되는 유전자의 mRNA에 혼성화할 수 있는 핵산 분자 (프로브)와 접촉시키는 것을 수반한다. mRNA는 고체 표면 위에 고정되고, 그리고 예를 들면, 단리된 mRNA를 아가로오스 겔 위에 이동시키고 mRNA을 겔로부터 막, 예컨대 니트로셀룰로오스로 이전함으로써 프로브와 접촉될 수 있다. 프로브(들) 역시 고체 표면 상에 고정될 수 있고, 그리고 mRNA가 예를 들면, AFFYMETRIX 유전자 칩 어레이 내에서 프로브(들)와 접촉된다. 당해 분야에서 공지된 mRNA 검출 방법이 관심되는 유전자의 mRNA의 수준을 결정하는 데 이용하기 위해 적합될 수 있다.
샘플 내에 관심되는 유전자의 발현 수준을 결정하기 위한 대안적 방법은 예를 들면, RT-PCR (Mullis, 1987, U.S. 특허 번호 4,683,202에서 진술된 실험 구체예), 리가아제 연쇄 반응 (Barany (1991) Proc. Natl. Acad. Sci. USA 88:189-193), 자가 지속 서열 복제 (Guatelli et al. (1990) Proc. Natl. Acad. Sci. USA 87:1874-1878), 전사 증폭 시스템 (Kwoh et al. (1989) Proc. Natl. Acad. Sci. USA 86:1173-1177), Q-베타 복제효소 (Lizardi et al. (1988) Bio/Technology 6:1197), 회전 원형 복제 (Lizardi et al., U.S. 특허 번호 5,854,033) 또는 임의의 다른 핵산 증폭 방법에 의한, 예를 들면 샘플 내에 mRNA의 핵산 증폭 및/또는 역전사효소 (cDNA 제조), 그 이후에 당해 분야에서 널리 알려진 기술을 이용한, 증폭된 분자의 검출의 과정을 수반한다. 이들 검출 기법은 만약 이런 분자가 매우 적은 숫자로 존재하면, 핵산 분자의 검출에 특히 유용하다. 본원 발명의 특정한 양상에서, 관심되는 유전자의 발현 수준은 정량적 형광원 RT-PCR (다시 말하면, TAQMANTM 시스템) 또는 DUAL-GLO® 루시페라아제 검정에 의해 결정된다.
관심되는 유전자의 mRNA의 발현 수준은 막 블롯 (예컨대 혼성화 분석 예컨대 노던, 서던, 반점 등에서 이용됨), 또는 마이크로웰, 샘플 튜브, 겔, 비드 또는 섬유 (또는 결합된 핵산을 포함하는 임의의 고형 지지체)를 이용하여 모니터링될 수 있다. 본원에서 참조로서 편입되는 U.S. 특허 번호 5,770,722; 5,874,219; 5,744,305; 5,677,195; 및 5,445,934를 참조한다. 유전자 발현 수준의 결정은 또한, 핵산 프로브를 용해 상태에서 이용하는 것을 포함할 수 있다.
mRNA 발현의 수준은 또한, 분지된 DNA (bDNA) 검정 또는 실시간 PCR (qPCR)을 이용하여 사정될 수 있다. 이러한 PCR 방법의 이용은 본원에서 제시된 실시예에서 설명되고 예시된다. 이런 방법은 또한, 관심되는 유전자의 핵산의 검출에 이용될 수 있다.
게다가, Grik2 유전자의 발현에 의해 생산된 GluK2 단백질의 수준은 단백질 수준의 계측을 위한 당해 분야에서 공지된 임의의 방법을 이용하여 결정될 수 있다. 이런 방법은 예를 들면, 전기이동, 모세관 전기이동, 고성능 액체 크로마토그래피 (HPLC), 박층 크로마토그래피 (TLC), 과다확산 크로마토그래피, 유체 또는 겔 침전소 반응, 흡수 분광법, 비색 검정, 분광측량 검정, 유세포분석, 면역확산 (단일 또는 이중), 면역전기이동, 웨스턴 블로팅, 방사면역검정 (RIA), 효소 결합 면역흡착 검정 (ELISA), 면역형광 검정, 전기화학발광 검정 등을 포함한다. 이런 검정은 또한, 관심되는 유전자에 의해 생산된 단백질의 존재 또는 복제를 표시하는 단백질의 검출에 이용될 수 있다. 추가적으로, 상기 검정은 단백질 기능에서 회복 또는 변화를 야기하고, 그것에 의하여 치료 효과 및 유익성을 개체에게 제공하는, 개체에서 장애를 치료하는 및/또는 개체에서 장애의 증상을 감소시키는, 관심되는 mRNA 서열의 변화를 보고하는 데 이용될 수 있다.
따라서, Grik2 mRNA 또는 GluK2 단백질 발현을 계측하기 위한 전술된 검정은 개체 (예를 들면, 뇌전증, 예컨대 예를 들면, TLE를 겪는 개체)를, 본원에서 개시된 하나 이상의 ASO 작용제 (예를 들면, 표 2에서 설명된 ASO 작용제 중에서 하나) 또는 이것을 인코딩하는 핵산 벡터를 이용한 치료적 처치가 필요한 것으로 확인하는 데 이용될 수 있다. 예를 들면, TLE를 겪는 것으로 확인된 환자는 통제할 수 없는 (예를 들면, 치료 내성, 예를 들면, 만성) 발작을 야기하는, 뇌의 한쪽 반구의 측두엽 내에 뇌전증유발 병소를 나타낼 수 있다. 본원에서 논의된 바와 같이, 이런 뇌전증유발 병소는 예를 들면, 재발성 치상 과립 세포 이끼 섬유의 일탈적 발아 및 상기 이끼 섬유에 의해 형성된 재발성 시냅스에서 Grik2의 비정상적인 (다시 말하면, 증가된) 발현으로부터 발생할 수 있다. 전술된 검정을 이용하여, 예를 들면, 뇌전증유발성 반구의 해마로부터 수집된 뇌 조직 및 건강한 반구 내에 동일한 영역으로부터 뇌 조직에서 작은 생검을 수행함으로써, 개체가 본원에서 개시된 Grik2 ASO 작용제 중에서 하나 이상을 이용하는 요법으로부터 유익성을 얻을 것인지에 관한 결정이 이루어질 수 있다. 뇌전증유발성 반구가 영향을 받지 않은 반구와 비교하여 Grik2 mRNA 또는 GluK2 단백질의 더 높은 (예를 들면, 적어도 5%, 10%, 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과) 발현 수준을 나타낸다는 제시는 환자가 본원에서 개시된 방법과 조성물을 이용한 요법으로부터 유익성을 얻을 수 있다는 것을 표시할 것이다. TLE를 겪는 개체가 양쪽 뇌 반구에서 뇌전증유발 병소를 나타내는 경우에, Grik2 mRNA 또는 GluK2 단백질 수준은 상기 개시된 검정을 이용하여, TLE를 겪는 개체의 하나 이상의 반구로부터 획득된 해마 조직 및 건강한 대조 개체의 동일한 반구(들)로부터 (예를 들면, TLE가 없는 개체의 사후 조직으로부터) 획득된 해마 조직 사이에 비교될 수 있었다. TLE를 겪는 개체의 뇌전증유발성 반구(들)가 건강한 개체의 동일한 반구(들)와 비교하여 Grik2 mRNA 또는 GluK2 단백질의 더 높은 (예를 들면, 적어도 5%, 10%, 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과) 발현을 나타낸다는 제시는 TLE를 겪는 개체가 개시된 조성물과 방법을 이용한 치료적 처치로부터 유익성을 얻을 것이라는 것을 표시할 것이다. 치료가 필요한 것으로 의심되는 개체의 뉴런 세포에서 Grik2 mRNA 수준 또는 GluK2 단백질 수준은 또한, 질환 상태를 표시하는 것으로 알려져 있는, 이들 피분석물의 표준 또는 참조 수준과 비교될 수 있다.
이에 더하여, 전술된 검정은 개체 (예를 들면, 뇌전증, 예컨대 예를 들면, TLE를 겪는 개체)가 본원에서 개시된 조성물과 방법을 이용한 치료에 반응했는지를 결정하는 데 활용될 수 있다. 예를 들면, 상기 논의된 바와 같이, 본원에서 개시된 조성물과 방법을 이용한 치료에 앞서, 뇌전증유발성 뇌 반구(들)로부터 해마 뇌 조직이 TLE를 겪는 개체로부터 작은 생검에 의하여 획득될 수 있고, 그리고 Grik2 mRNA 또는 GluK2 단백질의 발현이 전술된 검정을 이용하여 사정될 수 있다. 개체는 이후, 본원에서 개시된 방법과 조성물에 따른 치료가 시행될 수 있다. 개시된 방법과 조성물을 이용한 치료 이후에 (예를 들면, 치료 후 1, 5, 10, 15, 30, 60, 90일, 또는 그 초과 시점에) 환자의 회복 다음에, 사정된 동일한 뇌 영역에서 두 번째 생검이 치료에 앞서 수행될 수 있고, 그리고 Grik2 mRNA 또는 GluK2 단백질의 수준이 다시 한 번 사정될 수 있다. TLE를 겪는 개체가 Grik2 mRNA 또는 GluK2 단백질의 더 낮은 (예를 들면, 적어도 5%, 10%, 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과) 발현 수준을 나타낸다는 제시는 개체가 치료에 반응성이었다는 것을 표시할 것이다. 대안으로, Grik2 mRNA 또는 GluK2 단백질 수준은 한 명 이상의 건강한 대조 개체로부터 그것의 발현에 대하여 비교될 수 있다. 치료 후 TLE를 겪는 환자에서 Grik2 mRNA 또는 GluK2 단백질 수준이 한 명 이상의 건강한 대조 개체에서 그것의 수준과 통계학적으로 구별하기 어렵다는 제시는 환자가 치료에 반응성이라는 것을 표시할 것이다. 치료를 받은 개체의 뉴런 세포에서 Grik2 mRNA 수준 또는 GluK2 단백질 수준은 또한, 질환 상태의 부재를 표시하는 것으로 알려져 있는, 이들 피분석물의 표준 또는 참조 수준과 비교될 수 있다.
치료 방법
뇌전증 (예를 들면, TLE)을 겪는 개체는 본원에서 설명된 조성물과 방법을 이용하여 치료될 수 있다. 조성물 (예를 들면, ASO 작용제 내포 조성물 또는 이것을 내포하는 벡터)은 치료가 필요한 개체 (예를 들면, 뇌전증 (예를 들면, TLE)을 겪는 것으로 진단되거나 또는 겪을 위험에 처해 있는 개체에게 예방적 처치로서 투여될 수 있다. 뇌전증이 발달할 위험에 처해 있는 개체는 치료가 시행될 때 뇌전증의 초기 증상을 보여줄 수 있거나 또는 아직 증상을 보여주지 않을 수 있다.
투여 루트
본원에서 개시된 조성물은 표준 방법을 이용하여, 개체 (예를 들면, TLE를 겪는 것으로 확인된 개체)에게 투여될 수 있다. 예를 들면, 본원에서 개시된 조성물은 예를 들면, 전신 투여를 비롯한 다수의 상이한 루트 중 임의의 것에 의해 투여될 수 있다. 전신 투여의 무제한적 실례는 경장 (예를 들면, 경구) 또는 비경구 (예를 들면, 정맥내, 동맥내, 경점막, 복막내, 피부외층, 점막내 (예를 들면, 비내 또는 설하), 근육내, 또는 경피) 투여를 포함한다. 추가의 투여 루트는 피내, 피하 및 경피 주사를 포함할 수 있다.
본원에서 개시된 조성물은 또한, ASO 작용제 또는 이것을 인코딩하는 핵산 벡터의 국부 전달에 적합한 방법을 이용하여 투여될 수 있다. 국부 투여의 무제한적 실례는 피부외층 (예를 들면, 국소), 관절내 및 흡입 루트를 포함한다. 특히, 개시된 조성물은 개체의 뇌 조직 (예를 들면, 신경 세포, 예컨대 예를 들면, 뉴런 및/또는 성상세포))에 국지적으로 투여될 수 있다.
특히, ASO 작용제 및 이것을 인코딩하는 핵산 벡터는 개체의 뇌 조직, 예컨대 증가된 뇌전증모양 활성을 나타내는 것으로 결정된 뇌 조직에 국지적으로 투여될 수 있다. 뇌에 국부 투여는 일반적으로, 시냅시스로 연결된 선택된 세포 모집단의 적어도 일부 세포가 조성물과 접촉되도록, ASO 작용제 또는 이것을 인코딩하는 핵 벡터의 뇌 세포 (예를 들면, 신경 세포)로의 전달에 적합한 임의의 방법을 포함한다. 벡터는 뉴런, 성상세포, 또는 둘 모두를 비롯한, CNS의 임의의 세포로 전달될 수 있다. 일반적으로, 벡터는 예를 들면, 척수, 뇌간 (수질, 뇌교 및 중뇌), 소뇌, 간뇌 (예를 들면, 시상 및 시상하부), 종뇌 (선조체, 대뇌 피질 (예를 들면, 후두엽, 측두엽, 두정엽 또는 전두엽 내에 피질 영역), 또는 이들의 조합의 세포를 비롯한 CNS의 세포, 또는 그 안에 세포의 임의의 적합한 하위모집단으로 전달된다. 전달을 위한 추가 부위는 적핵, 편도, 내후각 피질, 그리고 시상의 복외측 또는 전위 핵 내에 뉴런을 포함한다.
본원 발명의 벡터는 정위 주사 또는 미세주입에 의하여, CNS의 실질 또는 뇌실 내로 직접적으로 전달될 수 있다. 특정 실례에서, 본원 발명의 벡터는 개체의 뇌 내에 하나 이상의 뇌전증 병소에 직접적으로 전달될 수 있다. 예를 들면, 개체는 본원 발명의 벡터가 정위 주사에 의하여, 이종피질 (예를 들면, 해마) 또는 신피질 (예를 들면, 전두엽)의 어느 한쪽 또는 양쪽 반구 내로 직접적으로 투여될 수 있다. 특정 실례에서, 개체는 본원 발명의 벡터가 정위 주사에 의하여 해마의 어느 한쪽 또는 양쪽 반구 내로 직접적으로 투여된다. 대안으로, 본원 발명의 벡터는 예를 들면, AAV9 또는 AAVrh10을 포함하지만 이들에 한정되지 않는, CNS 조직에 대한 향성을 나타내는 벡터의 맥락에서, 정맥내 주사에 의해 투여될 수 있다.
본원 발명의 벡터를 특정 영역 및 CNS 세포의 특정 모집단에 특이적으로 전달하기 위해, 벡터가 정위 미세주입에 의해 투여될 수 있다. 예를 들면, 개체는 제자리에 외과적으로 고정된 (두개골 내로 나사로 고정된) 정위 프레임 기부를 가질 수 있다. 정위 프레임 기부 (예를 들면, 기준 마킹이 있는 MRI 양립성 정위 프레임 기부)를 갖는 뇌는 고해상 MRI를 이용하여 영상화된다. MRI 이미지가 이후, 정위 소프트웨어를 실행하는 컴퓨터로 전달된다. 일련의 두정, 시상 및 축면 영상이 본원 발명의 조성물을 뇌 내로 주사하는 데 이용된 삽관 또는 주사 바늘의 표적 주사 부위 및 궤적을 결정하는 데 이용된다. 상기 소프트웨어는 궤적을, 정위 프레임에 적합한 3차원 좌표로 직접적으로 번역한다. 유입 부위 위에 홀이 천공되고, 그리고 정위 기구가 배치되며, 주사 바늘이 소정의 깊이로 이식된다. 조성물 (예컨대 본원에서 개시된 조성물)이 표적 부위에 주사될 수 있다. 조성물이 바이러스 입자를 생산하기보다는, 통합 벡터를 포함하는 경우에, 벡터의 확산은 부수적이고 주로 주사 부위로부터 수동 확산의 함수이다. 확산의 정도는 벡터 대 유체 운반체의 비율을 조정함으로써 제어될 수 있다.
추가의 투여 루트는 직접적인 가시화하에 벡터의 국부 적용, 예를 들면, 표재성 피질 적용, 또는 다른 비-정위적 적용을 또한 포함할 수 있다. 벡터는 척수강내 (예를 들면, 대수조 내로 직접적으로), 뇌실 내 (예를 들면, 뇌실내 (ICV) 주사를 이용하여), 또는 정맥내 주사에 의해 전달될 수 있다.
한 가지 실례에서, 본원 발명의 방법은 정위 주사를 통한 뇌내 또는 뇌실내 투여를 포함한다. 하지만, 다른 공지된 전달 방법 또한, 본원 발명에 따라서 적합될 수 있다. 예를 들면, CNS의 전역에서 조성물의 더 광범위한 분포를 위해, 이것은 예를 들면, 요추 천자에 의해 뇌척수액 내로 주사될 수 있다. 조성물을 말초 신경계로 지향시키기 위해, 이것은 관심되는 신체 부위의 척수 내로, 하나 이상의 말초 신경절 내로, 또는 피부 아래에 (피하 또는 근육내) 주사될 수 있다. 일정한 상황에서, 조성물은 혈관내 접근방식을 통해 투여될 수 있다. 예를 들면, 조성물은 혈액 뇌 장벽이 교란되거나 또는 교란되지 않는 상황에서 동맥내 (경동맥) 투여될 수 있다. 게다가, 더 전역의 전달을 위해, 조성물은 만니톨을 포함하는 고장성 용액의 주입에 의해 달성된, 혈액 뇌 장벽의 "개방" 동안 투여될 수 있다.
소정의 사례에서 가장 적합한 투여 루트는 투여된 특정 조성물, 개체, 치료되는 특정 뇌전증, 약학적 조제 방법, 투여 방법 (예를 들면, 투여 시간 및 투여 루트), 개체의 연령, 체중, 성별, 치료되는 질환의 심각도, 개체의 식이, 그리고 개체의 배출 속도에 좌우될 것이다.
병용 요법
본원에서 개시된 조성물은 다른 치료제 또는 물리적 개입 (예를 들면, 재활 요법 또는 외과적 개입)을 비롯한, 한 가지 이상의 추가 치료 양상 (예를 들면, 1가지, 2가지, 3가지, 또는 그 초과의 추가 치료 양상)과 병용으로 뇌전증 (예를 들면, TLE)을 치료하기 위해 치료가 필요한 개체 (예를 들면, 인간 개체)에게 투여될 수 있다. 2가지 이상의 작용제가 동시에 투여될 수 있다 (예를 들면, 모든 작용제의 투여가 15 분, 10 분, 5 분, 2 분 또는 그 미만 이내에 일어난다). 이들 작용제는 또한, 공동조제를 통해 동시에 투여될 수 있다. 2가지 이상의 작용제는 또한, 2가지 이상의 작용제의 작용이 중첩되도록 순차적으로 투여될 수 있고, 그리고 이들의 병용 효과는 장애에 관련된 증상 또는 다른 파라미터에서 감소가 1가지 작용제, 또는 단독으로 또는 다른 작용제의 부재에서 전달된 치료로 관찰될 것보다 크게 하는 정도이다. 2가지 이상의 치료의 효과는 부분적으로 부가, 온전히 부가, 또는 부가 초과 (예를 들면, 상승적)일 수 있다. 각 치료제의 순차적 또는 실제적으로 동시 투여는 경구 루트, 정맥내 루트, 근육내 루트, 국부 루트, 그리고 점막 조직을 통한 직접적인 흡수를 포함하지만 이들에 한정되지 않는 임의의 적합한 루트에 의해 수행될 수 있다. 이들 치료제는 동일한 루트에 의해 또는 상이한 루트에 의해 투여될 수 있다. 예를 들면, 병용의 첫 번째 치료제는 정맥내 주사에 의해 투여될 수 있고, 반면 병용의 두 번째 치료제는 화합물 함침 마이크로카세트에서 국지적으로 투여될 수 있다. 첫 번째 치료제는 두 번째 치료제 전후에 즉시, 1 시간까지, 2 시간까지, 3 시간까지, 4 시간까지, 5 시간까지, 6 시간까지, 7 시간까지, 8 시간까지, 9 시간까지, 10 시간까지, 11 시간까지, 12 시간까지, 13 시간까지, 14 시간까지, 16 시간까지, 17 시간까지, 18 시간까지, 19 시간까지, 20 시간까지, 21 시간까지, 22 시간까지, 23 시간까지, 24 시간까지, 또는 1-7, 1-14, 1-21 또는 1-30 일까지 투여될 수 있다.
개체가 뇌전증 (예를 들면, TLE)을 겪는 것으로 진단되거나 또는 뇌전증이 발달할 위험에 처해 있는 경우에, 두 번째 치료제는 발프로에이트, 라모트리긴, 에토숙시이미드, 토피라메이트, 라코사미드, 레베티라세탐, 클로바잠, 스티리펜톨, 벤조디아제핀, 페니토인, 카르바마제핀, 프리미돈, 페노바르비탈, 가바펜틴, 프레가발린, 티아가빈, 조니사미드, 펠바메이트 및/또는 비가바트린을 포함하지만 이들에 한정되지 않는 한 가지 이상의 항뇌전증 약물 (AED)을 포함할 수 있다. 일부 경우에, 두 번째 치료 양상은 외과적 개입, 예컨대 예를 들면, 당해 분야에서 널리 알려진 방법, 예컨대 예를 들면, 방사선수술 (예를 들면, 감마 나이프 또는 레이저 절제)을 이용한 뇌전증유발 뇌 영역의 외과적 적출 (예를 들면, 측두엽 적출)일 수 있다. 본원 발명의 방법 및 조성물과 함께 시행될 수 있는 추가 치료 양상은 미주 신경 자극, 뇌심부 자극, 머리뼈경유 자성 자극, 그리고 케톤생성 식이를 포함한다.
특정 실례에서, 개체는 코르티코스테로이드 단독, 또는 타크로리무스 또는 라파마이신 (시로리무스)의 섭생, 예를 들면 미코페놀산과의 병용 또는 코르티코스테로이드 예컨대 프레드니솔론 및/또는 메틸프레드니솔론과의 병용을 비롯한, 면역 억제제가 투여될 수 있다. 당해 분야에서 널리 알려진 다른 면역 억제 섭생이 본원 발명의 방법 및 조성물과 함께 이용될 수 있다. 이런 면역 억제 치료는 유전자 요법 전, 유전자 요법 후, 또는 유전자 요법에 부수적으로 시행될 수 있다.
투약
본원에서 설명된 바와 같이 치료될 수 있는 개체는 뇌전증 (예를 들면, TLE)을 겪는 것으로 진단되거나 또는 뇌전증이 발달할 위험에 처해 있는 개체이다. 개시된 방법과 조성물을 이용하여 치료될 수 있는 개체는 예를 들면, 뇌전증의 치료에 관련된 한 가지 이상의 선행 치료적 개입을 경험한 개체 또는 뇌전증의 치료를 위한 선행 치료적 개입이 없는 개체이다.
본원 발명의 ASO 작용제는 다음 중에서 1가지 이상 (예를 들면, 2가지 이상, 3가지 이상, 4가지 이상)을 야기하는 데 효과적인 양에서 및 시간 동안 투여될 수 있다: (a) 개체의 세포에서 Grik2 mRNA 및/또는 GluK2 단백질의 수준 감소, (b) 장애의 개시 지연, (c) 개체의 생존 증가, (d) 개체의 진행 없는 생존 증가, (e) GluK2 단백질 기능의 회복 또는 변화, (f) 발작 재발의 위험 감소; (g) CNS에서 흥분독성 및 연관된 뉴런 세포 사멸의 감소; (h) CNS의 영향을 받은 영역 (예를 들면, 해마)에서 생리학적 흥분 억제 균형의 복구; 및/또는 (i) 뇌전증의 1가지 이상의 증상 (예를 들면, 뇌전증 발작의 빈도, 지속 기간 또는 강도, 허약, 부재, 갑작스러운 착란, 언어를 이해하거나 생성하는 데 문제가 있음, 인지 장애, 손상된 가동성, 현기증, 또는 균형 또는 조화의 상실, 마비, 그리고 감정적 조절장애)의 감소.
따라서, 본원 발명은 치료가 필요한 개체에서 뇌전증 (예를 들면, TLE)을 치료하기 위한 방법에 관계하는데, 여기서 상기 방법은 개체에서 Grik2 mRNA에 특이적으로 결합하고 GluK2 단백질의 발현을 저해하는 저해성 RNA (예를 들면, ASO, 예컨대 예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA, 또는 shmiRNA)를 인코딩하는 올리고뉴클레오티드를 포함하는 벡터의 효과량을 투여하는 단계를 포함한다. 특히, 본원 발명은 치료가 필요한 개체에서 뇌전증을 치료하는 방법을 제공하는데, 상기 방법은 본원에서 개시된 ASO 작용제 또는 이것을 인코딩하는 핵산 벡터의 치료 효과량을 상기 개체에게 투여하는 단계를 포함한다.
개시된 방법과 조성물을 활용하여 치료되는 뇌전증은 TLE (예를 들면, mTLE 또는 lTLE), 양성 롤란드 뇌전증, 전두엽 뇌전증, 영아 연축, 청소년기 근간대 뇌전증, 청소년기 소발작 뇌전증, 아동기 소발작 뇌전증 (피크노렙시), 온수 뇌전증, 레녹스 가스타우트 증후군, 랜도 크레프너 증후군, 드라베 증후군, 진행성 근간대 뇌전증, 반사 뇌전증, 라스무센 증후군, 변연 뇌전증, 뇌전증 지속증, 복부 뇌전증, 거대 양측성 근간대경련, 월경 뇌전증, 작소니언 발작 장애, 라포라 증후군, 그리고 광민감 뇌전증일 수 있다. 예를 들면, 개체는 TLE (예를 들면, mTLE 또는 lTLE), 예컨대, 초점성 또는 전신 발작에 의해 특징화되는 TLE로 진단될 수 있다. 일부 경우에, 뇌전증은 만성 뇌전증, 예컨대 예를 들면, 치료에 불응성인 뇌전증 (다시 말하면, 약제 내성 뇌전증, 예컨대 약제 내성 TLE)일 수 있다.
뇌전증의 치료를 위해, 그리고 본원에서 논의된 바와 같은 발작 및 뇌전증모양 방전의 증상을 개선하기 위해, Grik2 mRNA의 발현을 저해하는 기능적 RNA, 예를 들면, siRNA, shRNA, miRNA, 또는 shmiRNA를 인코딩하는 벡터에 의해, 유용한 폴리뉴클레오티드가 배치될 수 있다.
개시된 조성물은 당업자에 의해 적합한 것으로 결정된 양으로 투여될 수 있다. 일부 경우에, rAAV는 개체마다 105, 106, 107, 108, 109, 1010, 1011, 1012, 1013, 1014, 또는 1015개 유전체 사본 (GC)의 용량으로 투여된다. 일부 구체예에서 rAAV는 105, 106, 107, 108, 109, 1010, 1011, 1012, 1013, 또는 1014개 GC/kg (개체의 전체 체중)의 용량으로 투여된다.
일부 경우에, 1 x 1012개 내지 5 x 1014개 GC가 투여된다. 일부 경우에, 1 x 1012개 내지 5 x 1014개 GC의 편평 용량이 소아 환자 또는 성인 환자에게 투여된다.
일부 경우에, 용량은 환자의 뇌 질량의 그램당 환자의 뇌척수액 (CSF)에 투여된 (예를 들면, 후두하 천자 또는 요추 천자를 통해, 예를 들면 척수강내 주사된) GC의 수에 의해 계측된다. 일부 경우에, 환자의 뇌 질량의 그램당 105, 106, 107, 108, 109, 109, 1010, 1011, 1012, 1013, 1014 또는 1015개 유전체 사본이 투여된다. 일부 경우에, 환자의 뇌 질량의 그램당 1 x 105개 유전체 사본이 투여된다. 일부 경우에, 환자의 뇌 질량의 그램당 1 x 106개 유전체 사본이 투여된다. 일부 경우에, 환자의 뇌 질량의 그램당 1 x 107개 유전체 사본이 투여된다. 일부 경우에, 환자의 뇌 질량의 그램당 1 x 108개 유전체 사본이 투여된다. 일부 경우에, 환자의 뇌 질량의 그램당 1 x 109개 유전체 사본이 투여된다. 일부 경우에 환자의 뇌 질량의 그램당 1 x 1010개 유전체 사본이 투여된다. 일부 경우에 환자의 뇌 질량의 그램당 5 x 1010개 유전체 사본이 투여된다. 일부 경우에 환자의 뇌 질량의 그램당 1 x 109개 내지 1 x 1011개 유전체 사본이 투여된다. 일부 경우에 환자의 뇌 질량의 그램당 1 x 109개 내지 5 x 1010개 유전체 사본이 투여된다. 일부 경우에 환자의 뇌 질량의 그램당 2 x 109개 내지 9 x 1010개 유전체 사본이 투여된다. 일부 경우에 환자의 뇌 질량의 그램당 5 x 109개 내지 1 x 1011개 유전체 사본이 투여된다. 다른 사례에서, 환자의 뇌 질량의 그램당 1 x 1010개 내지 5 x 1010개 유전체 사본이 투여된다. 다른 구체예에서, 환자의 뇌 질량의 그램당 1 x 1010개 내지 9 x 1010개 유전체 사본이 투여된다. 환자 (개체)의 뇌 중량 추정치는 MRI 뇌 용적 결정으로부터 획득되는데, 이것은 뇌 질량으로 전환되고 투여된 약물의 정확한 용량을 계산하는 데 활용된다. 뇌 중량은 또한, 공개된 데이터베이스를 이용하여, 연령 범위에 근거하여 추정될 수 있다.
임의적으로, 개시된 작용제는 본원에서 설명된 바와 같이, 개체에게 전달하기에 적합한 약학적으로 허용되는 조성물의 일부로서 투여될 수 있다. 개시된 작용제는 당업자에 의해 쉽게 결정될 수 있는 바와 같이, 원하는 용량을 제공하는 및/또는 치료적으로 유익한 효과를 이끌어 내는 데 충분한 양으로 이들 조성물 내에 포함된다.
본원에서 설명된 개시된 조성물은 개체를 치료하거나 또는 전술된 결과 중에서 한 가지 (예를 들면, 개체에서 질환의 한 가지 이상의 증상에서 감소)를 달성하는 데 충분한 양 (예를 들면, 효과량)으로 및 시간 동안 투여될 수 있다. 개시된 조성물은 1회 또는 1회 초과하여 투여될 수 있다. 개시된 조성물은 하루 1회, 하루 2회, 하루 3회, 2 일마다 1회, 주 1회, 주 2회, 주 3회, 격주 1회, 월 1회, 격월 1회, 연 2회, 또는 연 1회 투여될 수 있다. 치료는 별개 (예를 들면, 주사) 또는 연속 (예를 들면, 이식물 또는 주입 펌프를 통한 치료)일 수 있다. 개체는 치료에 이용된 조성물 및 투여 루트에 따라서, 본원 발명의 조성물의 투여 이후에 1 주, 2 주, 1 개월, 2 개월, 3 개월, 4 개월, 5 개월, 6 개월 또는 그 초과 시점에 치료 효능에 대해 평가될 수 있다. 치료 효능을 평가하는 방법은 본원에서 개시된다 (예를 들면, "약학적 용도" 참조). 평가의 결과에 따라서, 치료가 지속되거나 또는 중단될 수 있고, 치료 빈도 또는 용량이 변화될 수 있고, 또는 환자가 상이한 개시된 조성물로 치료될 수 있다. 개체는 별개의 기간 (예를 들면, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 또는 12 개월) 동안 또는 질환 또는 장애가 경감될 때까지 치료될 수 있거나, 또는 치료는 치료되는 질환 또는 장애의 심각도 및 성격에 따라서 장기적일 수 있다. 예를 들면, TLE로 진단되고 본원에서 개시된 조성물로 치료된 개체는 만약 초기 또는 후속 라운드의 치료가 치료적 유익성 (예를 들면, 본원에서 개시된 증상 중에서 한 가지의 감소, 또는 개체의 고통받는 뇌 영역에서 Grik2 mRNA의 수준 또는 GluK2 단백질 수준에서 감소)을 이끌어 내지 못하면, 1회 이상 (예를 들면, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10회, 또는 그 초과)의 추가 치료가 제공될 수 있다.
키트
본원 발명은 또한, 뇌전증 (예를 들면, TLE, 예컨대 치료 불응성 TLE)의 예방 또는 치료에 이용하기 위한, 개체에서 Grik2 유전자의 발현을 저해하는 본원에서 개시된 조성물 (예를 들면, Grik2 mRNA를 표적으로 하는 ASO)을 포함하는 키트를 제공한다. 키트는 임의적으로, 조성물을 개체에게 전달하기 위한 작용제 또는 장치를 포함할 수 있다. 다른 실례에서, 키트는 하나 이상의 무균 살포기, 예컨대 주입기 또는 바늘을 포함할 수 있다. 게다가, 키트는 임의적으로, 다른 작용제, 예를 들면, 마취제 또는 항생제를 포함할 수 있다. 키트는 또한, 키트의 사용자, 예컨대 의사가 본원에서 개시된 방법을 수행하도록 하기 위한 설명서를 포함하는 포장 삽입물을 포함할 수 있다.
실시예
하기 실시예는 당업자에게 본원에서 설명된 조성물과 방법이 어떻게 이용되고, 만들어지고, 평가될 수 있는지에 관한 설명을 제공하기 위해 제안되고, 본원 발명을 전적으로 예시하는 것으로 의도되며, 본 발명자들이 그들의 개시로서 간주하는 것의 범위를 한정하는 것으로 의도되지 않는다.
실시예 1A:
Grik2
mRNA 표적화 안티센스 올리고뉴클레오티드 설계 및 선택
Grik2 mRNA 표적화 안티센스 올리고뉴클레오티드의 설계
Grik2 mRNA를 표적으로 하고 이것의 발현을 저해할 수 있는 안티센스 올리고뉴클레오티드 (ASO)를 개발하기 위해, Grik2 mRNA 서열의 수집물 (5' 비번역 영역 (UTR), 코딩 영역 (CDS), 그리고 3' UTR)이 호모 사피엔스 (Homo sapiens)뿐만 아니라 무스 무스쿨루스 (Mus musculus), 라투스 노르베기쿠스 (Rattus norvegicus) 및 마카카 물라타 (Macaca mulatta)를 비롯한 다른 동물 모형 종의 NCBI GenBank 데이터베이스로부터 획득되었다. 가장 긴 전사체 변이체 (다시 말하면, 서열 번호: 115)에 초점이 맞춰지긴 했지만, 다른 전사체 변이체가 수집물 내에 포함되었다. 평가된 서열은 서열 번호: 115 (NM_021956 (호모 사피엔스 (H. sapiens))), 서열 번호: 117 (NM_175768 (호모 사피엔스 (H. sapiens))), 서열 번호: 118 (NM_001166247 (호모 사피엔스 (H. sapiens))), 서열 번호: 119 (NM_001111268 (무스 무스쿨루스 (M. musculus))), 서열 번호: 120 (NM_010349 (무스 무스쿨루스 (M. musculus))), 서열 번호: 121 (NM_001358866 (무스 무스쿨루스 (M. musculus))), 서열 번호: 122 (XM_015136995 마카카 물라타 (M. mulatta)), 서열 번호: 123 (XM_015136997 마카카 물라타 (M. mulatta)), 서열 번호: 124 (NM_019309.2 라투스 노르베기쿠스 (R. norvegicus))를 포함한다. 종 사이에 유의미한 서열 상동성의 영역을 확인하기 위해 서열 정렬 프로그램 MUSCLE를 이용하여 서열 정렬이 수행되었고, 그리고 치료적 개발 과정 내내 관심되는 각 종에서 원하는 전사체를 효과적으로 표적화하는, 소정의 가이드 서열의 가능성을 증가시키기 위하여, 가이드 RNA (다시 말하면, Grik2 mRNA와의 상보성 염기 대합에 관련된 ASO 서열) 설계 우선권이 이들 영역에 부여되었다.
기존 문헌은 mRNA의 이차 구조가 RNA 간섭 (RNAi)에 의해 표적화의 효율에 대한 유의미한 효과를 가질 수 있다고 제안한다 - 이차 구조를 내포하지 않는 영역은 RNAi 기구의 접근성이 더 높고, 이런 이유로 더 효율적으로 표적화되는 것으로 가정된다. 반대로, 염기 대합에 참여하여, RNA-RNA 이중나선을 형성할 것으로 예측되는 mRNA의 영역은 RNAi 기구의 접근성이 더 낮아, RNAi 접근방식에 의해 덜 효율적으로 표적화되는 것으로 가정된다. Grik2 mRNA의 이차 구조는 임의의 종에 대해 특징화되지 않는다. 따라서, 접힘된 RNA의 자유 에너지 (다시 말하면, 최소 자유 에너지 구조, 또는 MFE)를 감소시키는 것 또는 모든 다른 가능한 접힘 배향 (중심)과 비교하여 염기쌍 거리를 감소시키는 것 중 어느 하나에 근거된 알고리즘을 이용하는 예측 소프트웨어를 이용하는 전략이 창안되었다. 이러한 사정은 RNAfold WebServer (rna.tbi.univie.ac.at//cgi-bin/RNAWebSuite/RNAfold.cgi)를 이용하여, 호모 사피엔스 (Homo sapiens), 무스 무스쿨루스 (Mus musculus) 및 마카카 물라타 (Macaca mulatta)로부터 유래된 Grik2 mRNA 변이체 1에서 수행되었다. 낮은 염기 대합 확률 및/또는 높은 위치 엔트로피를 갖는 각 전사체의 영역 (도 1a 상에서 황색에서 청색으로 착색됨)이 확인되었고 가이드 설계를 위해 우선적으로 처리되었다. 이들 영역은 명확하게 묘사된 루프 영역 (도 1b에서 아라비아 숫자 1-14로서 지정되며, 이들은 각각, 서열 번호: 145-158에 상응한다)뿐만 아니라, 스템 유사 영역으로서 묘사되지만 염기 대합의 확률이 낮은 영역 (도 1b에서 아라비아 숫자 15-19에 의해 지정되며, 이들은 각각, 서열 번호: 159-163에 상응한다)을 포함하였다. 하나 초과의 종에서 낮은 염기 대합 확률 및/또는 높은 위치 엔트로피를 가질 것으로 예측된 영역에 우선권이 부여되었다. Grik2 mRNA (서열 번호: 115)에 대한 다양한 확인된 RNAi 가이드 서열의 정렬은 도 1c-1g에서 도시된다.
이후, 이들 영역을 표적으로 하도록 설계된 작은 저해성 RNA가 다른 전사체를 또한 표적화하여, 비표적 유전자의 조절이상에 기초된 바람직하지 않은 표적 이탈 효과를 야기할 잠재력을 가질 가능성을 예측적으로 결정하기 위해, 상기 열거된 방법에 의해 선택된 Grik2 mRNA 내에 관심 영역이 인실리코 평가되었다. Grik2 전사체 상에서 특정 영역에 대하여 설계된 가이드 RNA에 의해 매개된 표적 이탈 효과의 확률을 예측적으로 평가하기 위해, 온라인 도구 siSPOTR(sispotr.icts.uiowa.edu/sispotr/index.html_에서 가용한 "siRNA Sequence Probability-of-Off-Targeting Reduction")이 이용되었다. 관심되는 전사체를 입력한 후, 이러한 프로그램은 siRNA 또는 shRNA가 다른 전사체를 표적으로 할 가능성을 최소화하면서, 관심되는 전사체를 표적화할 것으로 예측되는 siRNA 또는 shRNA (이들 중 어느 한 쪽이 합성 ASO로 전환되고 그와 같이 전달될 수 있었다)의 목록을 확인한다. siSPOTR은 또한, 제안된 작은 RNA 분자로부터 유래된 시드 서열 (예를 들면, 표적 DNA 또는 mRNA 서열, 예컨대 Grik2 mRNA 서열과의 상보성 염기 대합에 관련된 서열)이 또한, 내생적으로 발현된 공지된 마이크로RNA (miRNA)의 시드 서열에 상응하는지를 표시하는데, 이것은 후보 분자가 선천적 miRNA에 의해 정상적으로 조절된 경로를 오조절할 수 있다는 것을 암시하였다. 이들 후보는 회피되었다.
RNAfold WebServer의 이용의 경우와 마찬가지로, 호모 사피엔스 (Homo sapiens), 무스 무스쿨루스 (Mus musculus) 및 마카카 물라타 (Macaca mulatta)로부터 유래된 전사체 변이체 1이 인간과 뮤린 전사체 둘 모두에 대해 siSPOTR을 이용하여 평가되었다. 이것은 인간 세포 (예를 들면, 탈체 표적 검증을 위한 시험관내 또는 인간 적출을 위한 세포주) 또는 뮤린 세포 (예를 들면, 효능의 생체내 뮤린 모형) 중 어느 하나에서 mRNA를 표적으로 할 낮은 가능도를 가질 것으로 예측된 각 전사체로부터 유래된 작은 RNA 후보의 확인을 가능하게 하였다. 이것은 또한, 인간과 뮤린 세포 둘 모두에서 최소 표적 이탈 효과를 야기할 것으로 또한 예측된 3개의 투입 전사체 모두에서 그들이 표적으로 하는 서열에서 상동한 작은 RNA 후보의 확인을 가능하게 하였는데, 이것은 약물 개발의 과정 동안 다양한 모형 전체에 대하여 효능에서 변동 및 표적 이탈 프로필 둘 모두를 최소화하기 위해 특히 관심된다.
최종적으로, 루시페라아제 리포터 시스템에서 합성 RNA 가이드를 이용하여 후보 표적 서열을 선별검사하는 목적으로, 인간 유전체로부터 발현된 임의의 RNA가 제안된 표적에 유의미한 유사성 (예를 들면, >90%, 이후 >80%)을 갖고, 이런 이유로 Grik2에 대하여 설계된 가이드에 의해 표적화될 가능성을 가질지를 결정하기 위해, 상응하는 21 bp 표적 서열이 "다소간 유사한 서열"에 대해 BLASTn (blast.ncbi.nlm.nih.gov/Blast.cgi?PAGE_TYPE=BlastSearch에서 가용함)을 이용하여 평가되었다.
세포주 리포터 검정을 이용한, GluK2 안티센스 올리고뉴클레오티드의 시험관내 스크린
인간 Grik2 (전사체 변이체 1) mRNA에 대한 서열 (5'와 3' UTR 포함)이 NCBI GenBank (NM_021956.4; 서열 번호: 115)로부터 획득되고, 합성되고, 그리고 pmiR-GLO-hGluK2로 명명되는 pMIR-GLO (Promega, 카탈로그 번호 E1330) 이중 루시페라아제 리포터 플라스미드의 맥락에서 포스포글리세린산 키나아제 (PGK) 프로모터의 제어하에 반딧불이 루시페라아제의 3' UTR 내로 클로닝되었다. 이런 이유로, PGK 프로모터로부터 발현은 인간 Grik2 mRNA를 표적으로 하는 작은 RNA 가이드의 평가를 가능하게 하는 하이브리드 반딧불이 루시페라아제-인간 Grik2 mRNA를 생성하였으며, Grik2 mRNA 발현의 RNAi 매개 감소는 반딧불이 루시페라아제 (ffluc) 리포터 단백질의 감소를 또한 야기하였다. 루시페라아제는 당해 분야에서 널리 알려진 다양한 방법에 의해 정량될 수 있다. 이러한 리포터 플라스미드는 또한, SV40 프로모터의 제어하에 레닐라 루시페라아제 리포터를 인코딩하는데, 이것은 자신의 3' UTR 내에 어떤 관심되는 표적화 서열도 내포하지 않기 때문에, 발현이 정규화 대조로서 역할을 하였다. ffluc:레닐라 신호의 비율 (RLU로 표현됨)이 이후, 보고되었고, 그리고 무관한 작은 RNA 가이드 (음성 대조로서 이용됨)가 동일한 방식으로 평가될 때 획득된 비율과 비교되었다. Grik2 mRNA 녹다운의 결과는 이런 이유로, "% GluK2 녹다운" (% KD로서 또한 지칭됨)으로서 보고되었고, 그리고 GluK2 단백질 발현에 대한 임의의 비특이적 효과는 아래와 같이, 음성 대조로부터 획득된 비율에 상대적인 "% 잔류 발현"으로서 계측되었다.
여기에서, 5ng pmiR-GLO-hGluK2가 작은 RNA 가이드, 이의 특정한 경우에 있어서, Grik2 mRNA에 대하여 설계된 10 pg의 합성 siRNA 또는 무관한 음성 대조 ASO와 함께, WT 293T 세포의 96 웰 평판 내로 동시형질감염되었다. 추가의 대조로서, 그리고 ASO 그 자체의 형질감염이 단백질 발현에 대한 더 포괄적인, 전역 효과를 갖는지를 결정하기 위해, 루시페라아제의 3' UTR 내에 Grik2 mRNA 서열을 내포하지 않아서, ffluc-hGluK2 하이브리드 mRNA를 생산하지 않는 빈 pmiR-GLO 플라스미드와 함께, 각 ASO가 추가적으로 동시형질감염되었다 (별개의 웰에서). 형질감염 48 시간 후에, 세포가 용해되었고, 그리고 반딧불이 및 레닐라 루시페라아제 발현 둘 모두가 이중-Glo 루시페라아제 검정 시스템 (Promega)을 이용하여 결정되고 상대적 발광 단위 (RLU)로 보고되었다. 상기 논의된 바와 같이, pmiR-GLO-hGluK2 리포터와 조합으로, Grik2 mRNA에 대한 실험적 ASO를 제공받는 웰에서 생성된 ffluc:레닐라 루시페라아제의 비율이 계산되고, 무관한 음성 대조 siRNA를 제공받는 웰로부터 획득된 비율과 비교되고, 그리고 표적 mRNA, 이 경우에 있어서 ffluc-hGluK2의 "녹다운 퍼센트" (% KD)로서 보고되었다 (도 1h; 표 2). 이들 값은 어떤 작은 RNA 가이드가 Grik2 mRNA 서열을 효율적으로 표적화하는지를 결정하는 데 이용되었다. 추가적으로, pmiR-GLO-빈 리포터와 조합으로, Grik2에 대한 실험 ASO 작용제를 제공받는 웰에서 생성된 ffluc:레닐라 루시페라아제의 비율이 계산되고, 무관한 음성 대조 ASO를 제공받는 웰로부터 획득된 비율과 비교되고, 그리고 비표적화된 ffluc mRNA로부터 "잔류 ffluc 발현 퍼센트"로서 보고되었다 (도 1i). 이들 값은 비표적화된 ffluc를 단백질 발현에 대한 대용물로서 이용하여, 어떤 작은 RNA 가이드가 형질감염된 세포에서 단백질 발현에 대한 비특이적 효과를 갖는지를 결정하는 데 이용되었다. 이들 값은 ffluc-hGluK2 mRNA 발현을 유의미하게 감소시키지만 비표적화된 ffluc 발현을 비특이적 방식으로 간섭하지 않는 가이드를 확인하기 위해, 가이드 후보 각각에 대해 플로팅되었다 (도 1j). 이러한 플롯으로부터, 가이드 선택을 위한 컷오프, 다시 말하면, >66% GluK2 녹다운 및 >66% 잔류 비표적화된 ffluc 발현이 확립되었다 (그래프 상에서 점선).
실시예 1B: Grik2 표적:siRNA 이중나선의 열역학적 특성
RNA-RNA 상호작용 및 이들의 열역학적 특성은 아래의 표 10 및 11에서 묘사된 바와 같이, RNAup WebServer을 이용하여 예측되었다. 아래와 같은 열역학적 특성: 결합의 총 자유 에너지, 이중나선 형성으로부터 에너지, 그리고 표적 개방 에너지 (개방 에너지 Grik2 mRNA)를 RNAup에 의해 사정하기 위해, 19개 염기쌍 (bp) siRNA (GluK2 mRNA와 상동성의 19개 염기를 보유) 및 21개 염기쌍 Grik2 siRNA (GluK2 mRNA와 상동성의 19 염기를 갖는 21개 염기 가이드를 보유)가 조회되었고, 그리고 이들 계산은 호의적인 가이드 특성을 상관시키기 위해, 가이드 각각에 대한 녹다운 퍼센트 (% KD, 실시예 1A 참조) 및 GC%의 결정과 비교되었다. RNAup는 본질적으로 2가지 단계에서 RNA-RNA 상호작용의 열역학을 계산하였다. 먼저, 잠재적인 결합 부위가 비대합된 상태로 남아있을 확률 (예를 들면 표적 부위를 개방하는 데 필요한 자유 에너지)이 계산되고, 이후 이러한 "접근성"은 총 결합 에너지를 획득하기 위해 상호작용 에너지와 합산된다.
표 10: 19-bp 합성 RNA 가이드에 대한 열역학적 특성 계산
표 11: 21 bp 성숙 miRNA 가이드에 대한 열역학적 특성 계산
표 10 및 표 11에 대한 칼럼 정의:
% KD: 후보 siRNA와 함께 동시형질감염에 의해 달성된 루시페라아제-GluK2 mRNA (Grik2 mRNA, 변이체 1, 서열 번호: 122) 리포터 발현의 녹다운 퍼센트 (실시예 1A에서 설명된 검정에 의해 결정된 바와 같은 동등한 19 bp RNA 가이드에 대해); 2개의 별표 (**)를 갖는 세포 = 33% 미만 녹다운 (KD), 1개의 별표 (*)를 갖는 세포 = 33-66% KD, 그리고 별표가 없는 세포 = 66% 초과 KD.
%GC: 구아닌 및 시토신인, siRNA의 가이드 서열 내에 염기의 합산 백분율; 표 10: 2개의 별표 (**)를 갖는 세포 = 60% 초과 GC, 1개의 별표 (*)를 갖는 세포 = 50-60% GC, 그리고 별표가 없는 세포 = 50% 미만 GC; 표 11: 2개의 별표 (**)를 갖는 세포 = 55% 초과 GC, 1개의 별표 (*)를 갖는 세포 = 50-55% GC, 그리고 별표가 없는 세포 = 55% 미만 GC.
굵은 글씨체로 표시된 Grik2 표적 영역은 본원 발명의 확인된 루프 또는 비대합 영역을 의미한다.
결합의 총 자유 에너지 (kcal/mol): mRNA 상에서 표적 영역의 개방, 단일 가닥 가이드의 생성, 그리고 단일 가닥 mRNA 표적 서열에 단일 가닥 siRNA 가이드의 혼성화를 비롯한, Grik2 mRNA (서열 번호: 122) 상에서 상응하는 표적 서열에 혼성화하는 siRNA의 과정의 자유 에너지; 표 10: 2개의 별표 (**)를 갖는 세포 = -28 kcal/mol 미만 (최대 델타G; 가장 음성 값), 1개의 별표 (*)를 갖는 세포 = -23 내지 -24 kcal/mol, 그리고 별표가 없는 세포 = -24 kcal/mol (가장 낮은 델타G; 제로에 가장 가까움); 표 11: 2개의 별표 (**)를 갖는 세포 = -30.5 kcal/mol 미만 (최대 델타G; 가장 음성 값), 1개의 별표 (*)를 갖는 세포 = -30.5 내지 -27 kcal/mol, 그리고 별표가 없는 세포 = -27 kcal/mol (가장 낮은 델타G; 제로에 가장 가까움).
이중나선 형성으로부터 에너지 (kcal/mol): 단일 가닥 mRNA 표적 서열에 단일 가닥 siRNA 가이드의 혼성화의 자유 에너지; 표 10: 2개의 별표 (**)를 갖는 세포 = -38 kcal/mol 미만 (최대 델타G; 가장 음성 값), 1개의 별표 (*)를 갖는 세포 = -38 내지 -35 kcal/mol, 그리고 별표가 없는 세포 = -35 kcal/mol (가장 낮은 델타G; 제로에 가장 가까움); 표 11: 2개의 별표 (**)를 갖는 세포 = -41 kcal/mol 미만 (최대 델타G; 가장 음성 값), 1개의 별표 (*)를 갖는 세포 = -41 내지 -38 kcal/mol, 그리고 별표가 없는 세포 = -38 kcal/mol (가장 낮은 델타G; 제로에 가장 가까움).
Gluk2 mRNA의 개방 에너지 (kcal/mol) (표적 개방 에너지로서 또한 알려져 있음): 표적 위치에서 RNA 이차 구조를 분해하는 데 필요한 에너지 (인근 이차 구조의 분해, 또는 표적 서열과 이차 구조를 형성하는 원위 서열의 관여를 포함할 수 있다); 표 10: 2개의 별표 (**)를 갖는 세포 = 가장 높은 에너지 요구량 (>10 kcal/mol = 가장 양성/최소 호의적), 1개의 별표 (*)를 갖는 세포 = 중간 에너지 요구량 (7.5 내지 10 kcal/mol; 중간 정도로 호의적), 별표가 없는 세포 = 가장 낮은 에너지 요구량 (<7.5 kcal/mol; 제로에 가장 가까움/가장 호의적); 표 11: 2개의 별표 (**)를 갖는 세포 = 가장 높은 에너지 요구량 (>9.5 kcal/mol = 가장 양성/최소 호의적), 1개의 별표 (*)를 갖는 세포 = 중간 에너지 요구량 (8 내지 9.5 kcal/mol; 중간 정도로 호의적), 별표가 없는 세포 = 가장 낮은 에너지 요구량 (<8 kcal/mol; 제로에 가장 가까움/가장 호의적).
GluK2 mRNA의 개방 에너지, 이중나선 형성으로부터 에너지, 그리고 결합의 총 자유 에너지를 결정하기 위해, RNAup WebServer (rna.tbi.univie.ac.at//cgi-bin/RNAWebSuite/RNAup.cgi, ViennaRNA Web Services software suite의 일부)가 이용되었다 (Lorenz, R., Bernhart, S.H., Hner zu Siederdissen, C., Tafer, H., Flamm, C., Stadler, P.F., and Hofacker, Ivo L. ViennaRNA Package 2.0, Algorithms for Molecular Biology, 6:1 26, 2011, doi:10.1186/1748-7188-6-26). RNAup는 2개의 투입 RNA 각각을 접고 (RNAfold 서버, rna.tbi.univie.ac.at/cgi-빈/RNAWebSuite/RNAfold.cgi에 의해 수행된 바와 같이), 이후 접힘된 RNA 각각의 열역학에 기초하여, 투입 RNA의 혼성화에 관련된 다양한 열역학적 값을 계산한다. 이들은 어느 한쪽 RNA (예를 들면, 표적 RNA 및 siRNA) 상에서 이차 구조를 개방하는 데 필요한 에너지 (kcal/mol), 이중나선 형성 (하나의 접히지 않은 RNA와 다른 접히지 않은 RNA의 혼성화)으로부터 생성된 에너지뿐만 아니라, 각 RNA를 개방하는 데 필요한 에너지 및 혼성화 그 자체의 에너지 둘 모두를 고려하는 총 결합 에너지를 포함한다.
여기에서, RNAup는 시험관내 또는 생체내 중 어느 한 가지의 실험 세팅에서 siRNA 활성 (예를 들면, 녹다운의 효능)을 예측할 수 있는 역치를 결정하기 위해, 인간 Grik2 mRNA (서열 번호: 122)에 결합하는 siRNA 가이드 서열의 열역학적 파라미터를 계산하는 데 이용되었다. 결과는 19 bp 합성 RNA 가이드의 경우 표 10에서, 그리고 21 bp 성숙 miRNA 가이드의 경우 표 11에서 열거된다. 본 실시예의 목적으로, Grik2 mRNA 서열에 상동성/상보적인 21개 염기 siRNA 서열 (실시예 1A에서 설명된 바와 같이 시험관내 루시페라아제 리포터 검정에서 검사된 siRNA)의 19개 염기가 전장 인간 Grik2 mRNA에 대해 조회되었다. 이러한 접근방식이 siRNA 서열의 19개 염기에 대하여 취해지긴 했지만, 이것은 21 bp 성숙 miRNA에 대해 수행된 것처럼, 더 긴 ASO, shRNA, miRNA 또는 shmiRNA를 조회하는 데 또한 이용될 수 있는데, 그 이유는 접힘/중첩풀림 및 혼성화의 원리가 일반적으로 비슷한 것으로 고려되기 때문이다. 전술된 열역학적 파라미터는 기록되었고 (표 10 및 11 참조), 그리고 루시페라아제 리포터 검정에서 % 녹다운 (KD)과 그들의 상관 및 이런 이유로, 소정의 가이드 서열의 효능을 예측하는 그들의 능력에 대해 평가되었다.
표적 개방 에너지
후보 가이드 서열이 GluK2 mRNA에서 그들의 상응하는 표적 서열을 개방하는 데 필요할 것으로 예측된 에너지에 따라서 순위 평가될 때, 낮은 개방 에너지 요구량 (0 kcal/mol에 가장 가까움)을 갖는 영역을 표적으로 하는 가이드 및 리포터 녹다운에 대한 더 큰 능력을 갖는 것으로 결정된 가이드 사이에 명백한 상관이 있다. 표적 개방 에너지 범위는 0.88 내지 17.71 kcal/mol인데, 평균이 8.12 kcal/mol이고 중앙값이 7.41 kcal/mol이다; 리포터 발현을 >66% 녹다운시킨 siRNA는 더 낮은 표적 개방 에너지 (평균 = 7.12 kcal/mol, 중앙값 = 6.66 kcal/mol)을 갖는 경향이 있는 반면, 리포터 발현을 그 만큼 효율적으로 녹다운시키지 못하는 siRNA는 더 높은 표적 개방 에너지 (평균 = 8.74 kcal/mol, 중앙값 = 8.34 kcal/mol)를 가졌다. 양쪽 군에서 이상점 (예를 들면, G0: %KD = 83, 반면 개방 에너지 = 17.24 kcal/mol; 그리고 MT: %KD = 4, 반면 개방 에너지 = 1.9 kcal/mol)이 있긴 했지만, 리포터 발현을 66% 초과 수준에서 녹다운시키는 siRNA 가이드 사이에 더 낮은 개방 에너지를 갖는 표적의 명백한 강화가 있었다. 기계적으로, 중첩풀림을 위해 더 적은 에너지 투입을 필요로 하는 표적 서열이 중첩풀림에 더 순응하고, 이런 이유로 siRNA 결합에 대한 "접근성이 더 높은" 것으로 고려될 수 있다는 결론이 나온다. 게다가, 도 1b 및 표 4에서 도시된 바와 같이, 예측된 이차성 Grik2 루프 및 비대합된 구조의 강화가 있다. 예를 들면, Grik2 mRNA의 단일 가닥 영역 내에 특이적으로 혼성화하는 23개 이내 뉴클레오티드의 길이를 갖는 RNA 가이드는 적어도 <10 kcal/mol 및 일정한 사례에서 <7.5 kcal/mol의 호의적인 개방 에너지를 나타낸다. 도 1b에서 도시된 바와 같이, 예측된 이차성 Grik2 루프 및 비대합된 구조에 결합하는 본원에서 개시된 가이드를 비롯한, siRNA의 강화된 클러스터는 10 kcal/mol 미만의 표적 개방 에너지를 갖고 66% 초과 Gluk2 녹다운과 상관되었다. <7.5 kcal/mol의 표적 개방 에너지 예측값을 갖는 19 bp Grik2 표적화 가이드는 유효할 가능성이 더 높고; 반면 >10 kcal/mol의 표적 개방 에너지 예측값은 일부 경우에 덜 호의적이다. <8.0 kcal/mol의 표적 개방 에너지 예측값을 갖는 21 bp Grik2 표적화 가이드는 유효할 가능성이 더 높고; 반면 >9.5 kcal/mol의 표적 개방 에너지 예측값은 일부 구체예에서 덜 호의적이다 (도 1j 및 1k).
이중나선 형성의 에너지
이후, 이중나선 형성의 예측된 에너지를 결정하기 위해 동일한 Grik2 mRNA/siRNA 가이드 서열 쌍이 조회되었다. 더 높은 리포터 녹다운을 갖는 가이드 및 이중나선 형성의 더 높은 에너지 (제로에 더 가까움)를 갖는 가이드 사이에 명백한 상관이 있었다. 이중나선 형성의 에너지 범위는 -42.7 내지 -12.92 kcal/mol이었는데, 평균이 35.28 kcal/mol이고 중앙값이 -35.41 kcal/mol이었다. >66% KD를 갖는 가이드 siRNA 서열은 평균적으로, 이중나선 형성의 더 높은 에너지 (평균 = -33.5 kcal/mol, 중앙값 = -33.3 kcal/mol)를 갖는 반면, <66% KD를 갖는 것들은 평균적으로, 이중나선 형성의 더 낮은 에너지 (평균 = -36.4 kcal/mol, 중앙값 = -37.5 kcal/mol)를 가졌다. 이중나선 형성의 더 음성/더 낮은 에너지는 소정의 이중나선의 형성이, 이중나선 형성의 에너지가 더 높거나/제로에 더 가까운 이중나선의 형성보다 더 호의적이라는 것을 암시한다. 이런 이유로, 이중나선 형성의 호감도 및 siRNA 가이드 서열 녹다운 효율의 효율 사이에 역 상관이 있는데, 이것은 반직관적인 것으로 보일 수 있다. 하지만, 이것은 이중나선 형성의 에너지가 이중나선 형성보다는 이중나선 분리 (이의 역)의 호감도를 결정할 때 더 결정적이라는 것을 암시한다. 이러한 문맥에서, 우리는 이러한 값을 이중나선 안정성의 척도로서 지칭한다; 값이 더 적을수록, 이중나선이 더 안정된다. 만약 표적:siRNA 이중나선이 덜 안정되면, siRNA는 증가된 진행성으로 인해 녹다운 관점에서 더 효율적일 수 있다. 다시 말하면, 효율과 직접적으로 관계하는, 상이한 mRNA 분자 상에서 동일한 영역을 표적화하기 위해, 이것은 덜 안정된 이중나선에서 떨어져 나올 가능성이 더 높다. 반대로, 만약 표적:siRNA 이중나선이 너무 안정되면, siRNA (RISC에 결합됨)는 표적에서 떨어져 나올 가능성이 더 낮다.
> -35 kcal/mol의 이중나선 형성 예측값의 에너지를 갖는 19 bp Grik2 표적화 가이드는 유효할 가능성이 더 높고; 반면 > -39 kcal/mol의 이중나선 형성 예측값의 에너지는 일부 구체예에서 덜 호의적이다. > -38 kcal/mol의 이중나선 형성 예측값의 에너지를 갖는 21 bp Grik2 표적화 가이드는 유효할 가능성이 더 높고; 반면 > -41 kcal/mol의 이중나선 형성 예측값의 에너지는 일부 구체예에서 덜 호의적이다 (도 1l 및 1m).
총 결합 에너지
최종적으로, 표적 GluK2 mRNA에 대한 siRNA/miRNA 가이드 서열 각각의 총 결합 에너지가 결정되었다. 19 bp siRNA 가이드의 경우에 총 결합 에너지 범위는 -35.06 내지 -9.89 kcal/mol이었는데, 평균이 -25.93 kcal/mol이고 중앙값이 -26.29 kcal/mol이었다. >66% KD 또는 <66%를 갖는 siRNA에 대한 총 결합 에너지에 대한 평균값은 현저하게 상이하지 않지만 (-25.65 대 -26.25 kcal/mol), 중앙값에서 차이는 더 컸는데 (각각, -25.35 대 -26.66 kcal/mol), >66% KD를 갖는 siRNA 가이드 중에서 -30 kcal/mol 미만의 총 결합 에너지를 갖는 것은 거의 없었다 (61개의 가이드 중 13개가 -30 kcal/mol 미만의 총 결합 에너지를 갖는 <66% KD 가이드와 비교하여, 37개 중 1개). 이런 이유로, 이것은 -30 kcal/mol 미만의 총 결합 에너지가 일부 경우에, 소정의 가이드 서열이 표적 mRNA를 효율적으로 녹다운시킬 가능성이 더 높다는 것을 표시한다는 것을 암시한다.
>-24 kcal/mol의 총 결합 에너지 예측값을 갖는 19 bp Grik2 표적화 가이드는 유효할 가능성이 더 높고; 반면 > -28 kcal/mol의 총 결합 에너지 예측값은 일부 구체예에서 덜 호의적이다. > -27 kcal/mol의 총 결합 에너지 예측값을 갖는 21 bp Grik2 표적화 가이드는 유효할 가능성이 더 높고; 반면 > -30.5 kcal/mol의 총 결합 에너지 예측값은 일부 구체예에서 덜 호의적이다 (도 1n 및 1o).
구아닌-시토신 (GC) 함량 (% GC)
최종적으로, siRNA 또는 miRNA 가이드 서열의 GC 함량이 결정되었다. 리포터 검정에서 >66% KD를 야기하는 siRNA 가이드의 군은 <66% KD를 갖는 나머지 가이드 (평균 = 56.4%, 중앙값 = 57.9%)보다 평균적으로 더 낮은 GC 함량 (평균 = 46.23%, 중앙값 = 47.4%)을 가졌다. 이런 이유로, 낮은 GC 함량은 가이드 효율의 강한 예측자이고, 그리고 반대로, 높은 GC 함량은 가이드 효율에 대한 대조지표이다. 이러한 상관에 기여하는 인자는 아마도 복합적이고 다수의 열역학적 파라미터에 관계한다. 먼저, 가이드 서열의 낮은 GC 함량은 또한, siRNA 이중나선 (또는, 전구체 shRNA 또는 miRNA 스템)의 승객 서열이 낮은 백분율의 GC를 내포한다는 것을 암시한다; 이런 이유로, 이러한 이중나선은 가이드 및 승객 가닥으로 더 쉽게 분리되어, 가이드 가닥이 A:U 대합으로부터 "더 약한" 염기 대합 기여의 강화로 인해 "성숙"할 수 있도록 한다. mRNA 내에 표적 서열은 이것을 표적으로 하는 가이드 서열에 관련된다; 이런 이유로, 만약 소정의 가이드가 낮은 GC 함량을 가지면, 표적 서열은 비록 완벽하게 상보적이지 않더라도, 아마도 낮은 GC 함량을 또한 갖는다. 만약 유의미한 이차 구조가 표적 서열의 부위에서 예측되면, 안티센스 RNA가 접근하도록 하기 위해 "개방"하는 데 더 적은 에너지가 필요할 것인데, 이것 역시 siRNA 표적화에 대한 호의적인 파라미터인 것으로 결정되었다. siRNA:표적 이중나선에서 더 낮은 GC 함량은 또한, 다시 한 번 더 안정된 G:C 염기 대합의 결여로 인한 이중나선의 더 낮은 안정성 (이중나선 형성의 에너지에 의해 계측될 때)을 야기하여 안티센스 RNA의 분리를 허용함으로써, 추가의 mRNA 분자가 표적화될 수 있도록 한다. 종합하면, siRNA의 GC 함량은 여기에서 설명된 다른 열역학적 파라미터에 직접적으로 관계하고, 그리고 상기 확립된 상관과 불가분의 관계에 있을 수 있다.
<50%의 GC 함량 값을 갖는 19 bp Grik2 표적화 가이드는 유효할 가능성이 더 높고; 반면 > 60%의 GC 함량 값은 일부 구체예에서 덜 호의적이다. <50%의 GC 함량 값을 갖는 21 bp Grik2 표적화 가이드는 유효할 가능성이 더 높고; 반면 > 55%의 GC 함량 값은 일부 구체예에서 덜 호의적이다 (도 1p 및 1q).
실시예 2: 뮤린, 설치류 및 인간 모형 시스템에서
Grik2
표적화
안티센스 올리고뉴클레오티드 작제물의 항뇌전증 효과의 검증
재료와 방법
생쥐를 이용한 프로토콜을 위해, 실험이 유럽 의회 지침 (2010/63/UE)에 따라서, Institut National de la Sant et de la Recherche Mdicale (INSERM) 동물실험윤리위원회에 의해 승인되고 Ministre de l'Education Nationale, de l'Enseignement Suprieur et de la Recherche에 의해 공인되었다.
뮤린 해마 기관형적 슬라이스
기관형적 해마 슬라이스 배양액 (350 μm)이 기존 문헌 (Peret et al. 2014)에서 설명된 바와 같이, McIlwain 조직 세단기를 이용하여 야생형 Swiss 생쥐 (P9-10)로부터 준비되었다. 슬라이스가 1 mL의 하기 배지: MEM 50%, HS 25%, HBSS 25%, HEPES 15 mM, 글루코오스 6.5 mg/mL 및 인슐린 0.1 mg/mL를 내포하는 배양 접시 내부의 그물망 삽입물 (Millipore) 상에 배치되었다. 배양 배지가 2 내지 3 일마다 교체되었고, 그리고 슬라이스가 37 ℃/5% CO2에서 인큐베이터 내에 유지되었다.
전기생리학적 기록
생쥐 기관형적 슬라이스가 30-32℃에 유지된 기록 챔버로 개별적으로 이전되고, 그리고 산소화된 (95% O2 및 5% CO2) ACSF (생리학적 상태), 또는 GABAA 수용체 길항제인 가바진을 내포하는 ACSF (5 μM; 과흥분 상태), 또는 5 μM 가바진 및 50 μM 4-AP를 내포하는 ACSF (고도로 과흥분 상태)로 연속적으로 관류되었다 (2-3 mL/분). 지역장 전위 (LFP) 기록이 DG의 과립 세포 층에 배치된 단극 니크롬선으로 이루어졌다. DAM-80 증폭기 (저역 필터: 0.1 Hz; 고역 필터: 3 KHz; World Precision Instruments, Sarasota, FL)가 이용되었다. 데이터가 Digidata 1440A (Molecular Devices)를 이용하여 컴퓨터로 디지털화되고 (20 kHz), 그리고 Clampex 10.1 소프트웨어 (PClamp, Molecular Devices)를 이용하여 획득되었다. 신호가 Clampfit 9.2 (PClamp) 및 MiniAnalysis 6.0.1 (Synaptosoft, Decatur, GA)을 이용하여 오프라인 분석되었다.
RNAi에 의한, Grik2 mRNA를 표적으로 하는 바이러스 벡터의 설계와 생산
RNAi (예를 들면, ASO) 서열이 Smart 선택 설계 (Dharmacon)(Birmingham et. al., 2007)를 이용하여 설계되었다. miR-30 구조를 이용한 shRNA 또는 miRNA 중 어느 하나로서 RNAi 서열의 효율이 비교되었다. CAG (서열 번호: 737) 프로모터 또는 hSyn 프로모터의 제어하에 인간 Grik2 안티센스 서열 (G9; 서열 번호: 68)을 인코딩하는 렌티바이러스 또는 AAV9 벡터가 이용되었다.
뉴런 세포 배양액
Gestant 암컷 쥐가 Janvier Labs (Saint-Berthevin, France)로부터 구입되었고, 그리고 야생형 생쥐가 생산되었다. 동물이 유럽 윤리 규범에 따라서 취급되고 안락사되었다. E18 Sprague-Dawley 쥐 배아로부터 분리된 해마 뉴런 또는 P0 생쥐로부터 분리된 피질 뉴런이 Kaech S. & Banker G (Culturing hippocampal neurons. Nat. Protoc. 1, 2406-2415 (2006))에 의해 설명된 바와 같이 준비되었다. 뉴런이 1 mg/mL 폴리리신으로 코팅된 6-웰 평판에 2 시간 동안 웰마다 500,000개 세포의 농도로 파종되었다. 뉴런이, 2 mM L-글루타민 및 1 NeuroCult SM1 뉴런 보충물 (STEMCELL Technologies)로 보충되고 3 내지 4 일마다 갱신된 조건화된 Neurobasal 배지 (쥐) 또는 Neurobasal-A 배지 (생쥐)에서 배양되었다. 도말 후 2 내지 3 일째에, 배지의 절반이 제거되었고, 그리고 바이러스 작제물이 4 시간 동안 MOI 75000로 첨가되었다. 감소된 배지 용적에서 바이러스 작제물과 4 시간의 접촉 후, 신경아교 세포 성장을 예방하기 위해, Ara-C (3.4 mM)로 보충된 새로운 배지가 첨가되었다.
웨스턴 블로팅
감염 후 10 일째에, DIV 12-13 뮤린 또는 쥐 뉴런 배양액이 얼음같이 차가운 PBS에서 헹굼되고, 이후 150 μL 용해 완충액 (50 mM HEPES, 100 mM NaCl, 1% 글리세롤, 0.5% n-도데실 β-D-말토시드, pH 7.2; 항-프로테아제 및 항-포스파타아제) 내로 긁어내졌다. 균질액이 차가운 온도에서 2 시간 동안 바퀴 상에 유지되었고, 그리고 세포 조직파편을 제거하기 위해 4 ℃에서 15 분 동안 8000g로 원심분리되었다. 전체 단백질 함량이 조건마다 10 μL에서 이중으로 Pierce BCA 단백질 검정 키트 (23225, ThermoScientific)에 의해 정량되었다.
조건마다 10 μg의 단백질이 웨스턴 블롯 분석을 위해 SDS-PAGE 겔 상에 부하되었다. 미리 염색된 단백질 사다리 (26619, ThermoScientific)가 중량을 제어하기 위해 부하되었다. 샘플이 4-15% 구배 프리캐스트 겔 (Bio-Rad)에서 분리되고, 이후 면역블로팅 분석을 위해 니트로셀룰로오스 막으로 이전되었다. 트리스-완충된 식염수 Tween-20 (TBST; 28 mM 트리스, 136.7 mM NaCl, 0.05% Tween-20, pH 7.4)에서 5% 소 혈청 알부민 (BSA; Sigma)으로 차단한 후, 샘플이 실온에서 1 시간 동안 셰이커 플랫폼 상에 유지되었다. 막이 72KDa 마커의 수준에서 사다리에 따라서 2 부분으로 절단되었다. 각 부분이 차단 시약 (37516, ThermoScientific; 항체 용액은 3회 재사용될 수 있었다)에서 희석된 개별 일차 항체와 함께 4℃에서 하룻밤 동안 인큐베이션되었다. 무거운 중량 단백질 (72-250 kDa)을 갖는 막 부분은 1:2000으로 희석된 토끼 항-GluK2 항체 (04-921; Merck)와 함께 인큐베이션되었고, 그리고 더 가벼운 중량 단백질 (17-72 kDa)을 갖는 막은 뮤린 항-β-액틴 항체 (A5316; Sigma)와 함께 인큐베이션되었다. 그 다음 날, 이들 막은 각각 TBST로 15 분 동안 3회 세척되었다. 당나귀에서 생산된 적합한 HRP-접합된 이차 항체 (Jackson ImmunoResearch)가 5% BSA-TBST에서 1:5000 희석되고 실온에서 1 시간 동안 막과 함께 인큐베이션되었다. 표적 단백질이 ChemiDoc Touch 시스템 (Bio-Rad)에서 clarity 웨스턴 ECL (170-5060, Bio-Rad)을 이용한 화학발광에 의해 검출되었다. GluK2의 이론적 분자량은 103 kDa이다. 정량을 위해, 각 레인의 화학발광 신호의 강도가 대조 조건 및 그 후에 액틴 띠에 대해 정규화되었다.
기관형적 슬라이스의 바이러스 형질도입
뮤린 슬라이스의 형질도입을 위해, 렌티바이러스 또는 AAV 작제물을 내포하는 1 μL의 PBS가 뮤린 슬라이스에 대한 DIV0에서 슬라이스 상에 직접적으로 떨어뜨려졌다. 바이러스 역가는 LV-대조의 경우 8x108개 유전체 사본 (GC)/mL, LV-G9-shRNA의 경우 7.9x108개 GC/mL, LV-G9-miRNA의 경우 3.0x109개 GC/mL, AAV9-G9-miRNA의 경우 2.8x1013개 GC/mL, 그리고 AAV9-GFP-GC의 경우 9.0x1012개 이었다.
면역표지화
Prox1 및 GFP 면역염색을 위해, 슬라이스가 고정되고, 이후 0.5% 트리톤에 5% 정상 염소 혈청 (NGS)을 내포하는 차단 용액에서 실온에서 1 시간 동안 투과화되었다 (0.5% 트리톤). 슬라이스는 이후, 5% NGS에서 1:2000으로 다중클론 토끼 항-Prox1 항체 (Millipore)와 함께, 그리고 0.5% 트리톤에서 1:1000으로 다중클론 닭 항-EGFP (Abcam, Cambridge, UK; RRID: AB-300798)와 함께 4 ℃에서 하룻밤 동안 인큐베이션되었다. 슬라이스가 이차 항체 Alexa488 (Invitrogen 1:500)에서 2 시간 동안 인큐베이션되고, 이후 Fluoromount (Thermo Fisher)에 커버슬립되었다. 형광 이미지가 10X/0.3NA 및 20X/0.8NA 대물렌즈를 이용한 LSM800 Zeiss 공초점 현미경으로 획득되었다. 이미지가 NIH ImageJ 소프트웨어를 이용하여 처리되었다.
통계학적 분석
모든 값은 별도로 특정되지 않으면 평균 ± SEM으로서 제공된다. 통계학적 분석이 Graphpad Prism 7 (GraphPad Software, La Jolla, CA)을 이용하여 수행되었다. 샤피로 윌크 검정이 데이터의 정규성을 결정하는 데 이용되었다. 파라미터 스튜던트 t 검정 (대응표본 및 독립표본, 양측)이 데이터의 정상적으로 분포된 집단을 비교하는 데 이용되었다. 만 휘트니 검정 (독립표본 데이터, 양측) 및 윌콕슨 부호 순위 검정 (대응표본 데이터)이 비-정상적으로 분포된 데이터에 이용되었다. 누적 분포의 비교를 위해, 콜모고로프 스미르노프 검정이 이용되었다. 복수의 집단의 비교를 위해, 일원 및 이원 변량 분석 검사가 이용되었다. 유의성 수준은 p<0.05로 설정되었다. 집단 척도는 평균 ± SEM으로서 표현된다; 오차 막대 역시 SEM을 표시한다.
결과
RNA 간섭에 의한 GluK2의 하향조절을 위한 바이러스 벡터의 설계와 검증
뇌 슬라이스에서 뇌전증모양 방전 (ED)의 생성에서 GluK2/GluK5 KAR의 특정한 역할을 더욱 확증하기 위해, GluK2/GluK5 내포 KAR의 수준을 하향조절하기 위한 바이러스 매개 RNA 간섭 (RNAi) 전략이 개발되었다.
인간 Grik2 mRNA에 대한 여러 가이드 RNA 서열이 Smart 선택 설계 (Birmingham et al. Nature Methods 9:2068-78, 2007)를 이용하여 설계되었다. 초기 실험이 뉴런에서 GluK2 발현을 잠재적으로 하향조절할 수 있는 하나의 RNAi 서열 (G9; 서열 번호: 68)의 확인을 가능하게 하였다. 이러한 RNAi 서열의 효능이 먼저, U6 프로모터의 제어하에 shRNA를 이용하여 사정되었고, 그리고 miRNA가 hSyn1 프로모터의 제어하에 인간 miR-30 구조를 이용하여 사정되었다. 이들 작제물은 LV 또는 AAV9 벡터 (도 2a-2d) 중 어느 하나에 의해 전달되었고, 그리고 웨스턴 면역블로팅에 의해 쥐 해마 뉴런에서 및 쥐 해마 뉴런에서 검사되었다. 이들 실험에서 이용된 렌티바이러스 및 AAV9 벡터의 실례는 아래의 표 12에서 설명된다:
표 12: 렌티바이러스 및 AAV9 벡터 작제물에 관한 설명
감염 후 10 일째에, GluK2 단백질의 총량이 액틴에 대해 정규화되고, 그리고 스크램블 대조 작제물 (LV: TTTGTGAGGGTCTGGTC (서열 번호: 771); AAV9: GC (서열 번호: 101)뿐만 아니라 AAV-CAG (서열 번호: 737)-EGFP 작제물과 비교하여 결정되었다. LV-hSyn (서열 번호: 682)-EGFP 작제물은 LV 벡터로 뉴런의 감염의 속도를 추정하는 것을 가능하게 하였는데, 이것은 뮤린 기관형적 슬라이스에서 대략 40-50%이었다 (도 2e). 세포에서 AAV 매개 바이러스 형질도입에 이용된 AAV 발현 카세트의 예시적인 계통도는 도 2g에서 도시된다. 안티센스 서열 G9 (서열 번호: 68)는 LV 벡터에서 및 AAV9 벡터에서 shRNA로서, miRNA로서 및 둘 모두로서, 총 GluK2 단백질의 수준을 감소시키는 데 효율적인 것으로 관찰되었다 (도 2f). 쥐 해마 배양액 (도시되지 않음)에서, 항-Grik2 mRNA 서열 (G9; 서열 번호: 68)을 인코딩하는 상이한 바이러스 작제물은 GluK2 단백질의 수준을 유의미하게 감소시키는 반면, LV-스크램블 (서열 번호: 771)은 어떤 유의미한 감소도 보여주지 않았다. 쥐 해마 뉴런 (도 2h)에서, 항-Grik2 서열 (G9; 서열 번호: 68)을 인코딩하는 상이한 바이러스 작제물은 GluK2 단백질의 수준을 유의미하게 감소시키는 반면, LV-스크램블 (서열 번호: 771)은 어떤 유의미한 감소도 보여주지 않았다.
항-Grik2 서열 (G9 (서열 번호: 68); GI (서열 번호: 77); XY (서열 번호: 83); Y9 (서열 번호: 88); GG (서열 번호: 91), 그리고 음성 대조 서열 (GC; UAAUGUUAGUCAUGUCCACcg; 서열 번호: 101)을 인코딩하는 여러 다른 바이러스 벡터가 뮤린 일차 피질 뉴런에서 GluK2 단백질 발현 (도 2i)을 녹다운시키는 능력에 대해 검사되었다. 배양된 피질 뉴런이 DIV3에서 7.5x104 감염 다중도로, 항-Grik2 서열을 인코딩하는 바이러스 벡터로 형질도입되었으며, 각 작제물이 삼중으로 검사되었다. 세포가 형질도입 후 10 일째에 용해되었다. 아래의 표 13은 도 2i에서 제시된 데이터에서 수행된 통계학적 분석의 요약을 제공한다. 모든 안티센스 작제물은 GluK2 단백질 수준에서 통계학적으로 유의한 감소를 나타내는 반면, 대조 서열은 유의미한 녹다운을 생성하지 않았다.
표 13. GluK2 단백질 발현을 녹다운시키는 다양한 항- Grik2 ASO 서열의 효능의 상대적인 통계학적 분석.
내생적으로 발현 세포에서 Grik2 mRNA의 발현을 평가하도록 의도된 실험의 별개 세트에서, 유도 만능 줄기 세포 (iPSC) 유래된 글루타민산성 뉴런 (iCell® GlutaNeurons - FCDI)이, 지질 기반 형질감염 시약 내로 통합된 hSyn 프로모터의 조절 제어하에 5가지 Grik2 mRNA 안티센스 올리고뉴클레오티드 (G9 (서열 번호: 68), GI (서열 번호: 77), Y9 (서열 번호: 88), XY (서열 번호: 83) 또는 MU (서열 번호: 96)), 또는 스크램블 대조 서열 (GC; 서열 번호: 101) 중에서 하나를 인코딩하는 플라스미드로 형질감염되었다. 형질감염 효율이 2:1 및 4:1 비율의 DNA : 지질 기반 형질감염 시약을 이용하여 비교되었다. TaqManTM 단일플렉스 실시간 정량적 중합효소 연쇄 반응 (RT-qPCR)이, 형질감염후 3개의 복제물을 이용하여 384-웰 평판 내에 GlutaNeurons에서 Grik2 녹다운의 수준을 정량하는 데 이용되었다. GlutaNeurons는 17,500개 세포/웰 (17.5k c/w)의 밀도로 도말되었다. Grik2 mRNA 발현이 GAPDH 신호에 대해 정규화되었다. Grik2 안티센스 작제물을 인코딩하는 플라스미드의 형질감염은 4:1의 DNA-대-지질 시약 비율로 G9 (서열 번호: 68), GI (서열 번호: 77) 및 Y9 (서열 번호: 88)를 내포하는 작제물에 경우에, 5 일 후 GlutaNeurons에서 Grik2 mRNA 발현을 유의미하게 감소시키는 것으로 밝혀졌다 (도 2j). 이들 조사 결과는 Grik2 유전자를 내생적으로 발현하는 세포에서 Grik2 mRNA의 발현을 녹다운시키는 Grik2 안티센스 올리고뉴클레오티드의 유용성을 증명한다.
GluK2 녹다운은 뮤린 기관형적 해마 슬라이스에서 뇌전증모양 활성을 억제한다
표적 세포에서 AAV9 작제물의 발현을 확증하기 위해, 프로스페로 호메오복스 1 (Prox1; Millipore) 및 녹색 형광 단백질 (GFP) 면역조직화학이, AAV9-스크램블-eGFP 벡터로 형질도입된 뮤린 해마 슬라이스에서 수행되었다. 실제로, Prox1 및 GFP의 광범위한 공동표지화가 DG 뉴런에서 관찰되었는데 (도 3a), 이것은 AAV9 벡터가 뮤린 DG 뉴런을 효율적으로 형질도입하고 관심되는 폴리뉴클레오티드를 발현할 수 있다는 것을 표시한다. 뮤린 해마 기관형적 슬라이스에서 기록된 ED의 주파수 (도 3b에서 도시된 예시적인 세포외 전압 트레이스)를 감소시키는, Grik2 안티센스 서열 (G9, 서열 번호: 68)을 인코딩하는 LV 또는 AAV 작제물의 shRNA 또는 miRNA로서의 효능이 기존 문헌 (Peret et al., 2014)에서 설명된 바와 같이, 과다흥분성을 생성하는 세포외 배지에서 검사되었다 (방법 참조). LV-hSyn (서열 번호: 682)--G9-miRNA (서열 번호: 68)은 대조 조건 (LV-hSyn (서열 번호: 682)-스크램블; 서열 번호: 771)과 비교하여 ED의 주파수를 강하게 감소시켰다 (LV-G9-miRNA의 경우 0.041 ± 0.006 Hz, n=24; LV-hSyn (서열 번호: 682)-스크램블 (서열 번호: 771)의 경우 0.066 ± 0.009 Hz, n= 12, p=0.0246, 만 휘트니 검정; 도 3c). 그 다음, AAV를 이용한 작제물이 검사되었는데, 그 이유는 이들 바이러스 벡터가 인간 유전자 요법을 위해 통상적으로 선택되기 때문이다. AAV9 혈청형이 이들 실험에서 이용되었다. AAV9-hSyn (서열 번호: 683)-G9 (서열 번호: 68)는 대조 조건 (AAV9-hSyn (서열 번호: 682)-GC; 서열 번호: 101)과 비교하여 ED의 주파수를 효율적으로 감소시켰다 (AAV9-G9-miRNA의 경우 0.025 ± 0.007 Hz, n=18; AAV9-GFP-GC의 경우 0.061 ± 0.007 Hz, n= 32, p=0.0004, 일원 변량 분석; 도 3c).
다양한 항-Grik2 ASO 서열을 인코딩하는 추가의 AAV9 작제물이 뮤린 기관형적 슬라이스에서 ED를 억제하는 능력에 대해 검사되었다. 간단히 말하면, 다양한 상이한 항-Grik2 ASO 서열을 인코딩하는 AAV9 벡터가, DIV0에서 9x109개 유전체 사본/mL의 바이러스 역가로 야생형 뮤린 기관형적 슬라이스에 형질도입되었다 (방법 참조). 5 μM 가바진을 내포하는 과흥분 배지에서 전기생리학적 기록이 DIV10-11에서 수행되었다. 무억제성 기관형적 슬라이스 준비물에서 생성된 ED의 저해가 검사된 5개 (5개 중) 작제물 (다시 말하면, G9 (서열 번호: 76), XY (서열 번호: 83), GI (서열 번호: 477), Y9 (서열 번호: 88) 및 GG (서열 번호: 91; 도 3d)에서 관찰되었다. 도 3d에 도시된 데이터의 통계학적 분석의 요약은 아래의 표 14에서 제공된다.
표 14: 뮤린 기관형적 뇌 슬라이스에서 뇌전증모양 활성을 억제하는, 항-
Grik2
RNA 서열을 인코딩하는 AAV9 벡터의 효능의 상대적인 통계학적 분석.
실시예 3: 측두엽 뇌전증의 뮤린 모형에서
Grik2
표적화 안티센스 작제물의 생체내 효능
방법과 재료
Grik2 표적화 ASO 작용제의 생체내 효능을 사정하기 위해, TLE의 필로카르핀 유발 모형이 hSyn 프로모터 (서열 번호: 682)의 제어하에 바이러스로 인코딩된 스크램블 서열 (GC; 서열 번호: 101), 또는 hSyn 프로모터 (서열 번호: 683)의 제어하에 바이러스로 인코딩된 항-Grik2 ASO 작용제 (G9; 서열 번호: 68)로 처리와 함께 생쥐에서 이용되었다. 0일 자 (D0)에, 뇌전증 지속증을 유도하기 위해 등면 (1 mL/반구) 및 복면 (1 mL/반구; 4 mL 총/생쥐) 내로, 필로카르핀의 양측성 해마내 주사가 생쥐에게 제공되었다. 생쥐는 이후, 해마 회로의 병리생리학적 재편성이 발생할 수 있도록 하기 위해 3-4 주가 부여되었다. 바이러스로 인코딩된 ASO 작용제로 주사 7 일 전 (D60)에, 생쥐에서 행동 사정이 진행되었다. 본원에서 논의된 바와 같이, TLE의 병인에 대한 신경해부학적 기질은 기억 및 학습에서 결정적인 역할로 인해 널리 알려진 뇌 영역인 해마이다. 학습 및 기억에 대한, Grik2 표적화 ASO 작용제를 해마 내로 투여하는 효과를 사정하기 위해, 10 마리의 필로카르핀 처리된 생쥐가 먼저, 새로운 물체 및 익숙한 물체를 인식하는 능력에 대해 새로운 물체 인식 (NOR) 과제에서 검사되었다. NOR 과제의 기본 구조는 첫 번째 세션 동안 생쥐에게 2개의 유사한 물체를 제시하고 생쥐가 이들 2개의 물체를 자유롭게 탐색하고 이들과 익숙해질 수 있도록 하는 것을 수반한다. 생쥐가 이들 물체에 익숙해짐에 따라서, 이들 이전에는 새로웠던 물체를 탐색하는 성향이 줄어든다. 두 번째 세션의 시작에 앞서, 이들 물체 중에서 하나가 새로운 물체에 의해 대체되고, 그리고 생쥐는 다시 한 번 양쪽 물체를 탐색하도록 허용된다. 일반적으로, 새로운 물체를 탐색할 때까지 잠재기는 인식 기억에 대한 프록시를 제공한다, 다시 말하면, 생쥐는 익숙한 물체보다, 새로운 물체를 탐색할 때까지 더 짧은 잠재기를 나타낼 것이다. 초기 행동 사정 후 7 일 (D67)째에, 생쥐는 hSyn 프로모터 (서열 번호: 682)의 제어하에 녹색 형광 단백질 (GFP)을 포함하는 스크램블 대조 서열 (GC; 서열 번호: 101) 또는 hSyn 프로모터 (서열 번호: 683)의 제어하에 Grik2 표적화 ASO 서열 (G9; 서열 번호: 68) 중 어느 하나를 인코딩하는 AAV 벡터의 9 x 1012개 유전체 사본/mL의 양측성 해마내 주사를 제공받았다. 전술된 ASO-인코딩 벡터에 관한 상세는 아래의 표 15에서 설명된다.
표 15: 연구에 이용된 안티센스 작제물에 관한 상세
바이러스 작제물을 이용한 주사를 제공받은 후 7 일 (D82)째에, 생쥐는 다시 한 번 NOR 과제가 진행되었다. 주사후 NOR 사정 후 7 일 (D89)째에, 생쥐는 뇌파검사법 (EEG) 전극이 해마내 이식되었고, 그리고 전자사진술 발작이 D96부터 D103까지 5 일 동안 기록되었다. 기록은 COVID-19으로 인해 더 이상 계속되지 않았다. 전자사진술 발작은 EEG 기준선의 적어도 2배의 EEG 진폭 및 적어도 6 초의 지속 기간을 갖는 발작성 사건으로서 규정되었다.
결과
TLE로 진단된 환자는 종종, 뇌전증 발작과 함께 다른 동시이환, 예컨대 예를 들면, 그 중에서도 특히, 학습 및 기억에서 결함을 나타낸다. 바이러스로 인코딩된, Grik2 표적화 안티센스 작제물의 투여에 의한 생체내에서 Grik2 발현의 저해를 결정하기 위해, NOR 과제 (도 4a)가 안티센스 작제물로 주사 전후에, 경련유발 필로카르핀으로 전처리된 생쥐에서 수행되었다. 검사 및 훈련 시기 동안 양쪽 물체를 탐색하는 데 소모된 시간 사이의 차이, 그리고 새로운 또는 익숙한 물체를 탐색하는 성향에 대한 프록시이기도 한 이동된 총 거리를 계측하는 식별 지수 (DI)에 의해 계측될 때, 스크램블 대조 서열 (GC; 서열 번호: 101)을 인코딩하는 AAV 벡터로 추후 처리되는 대조 생쥐는 항-Grik2 서열 (G9; 서열 번호: 76; 도 4b)을 인코딩하는 AAV 벡터로 추후 처리되는 생쥐와 유사한 성과를 나타냈다. 바이러스 벡터 주사 다음에, 항-Grik2 작제물로 처리된 생쥐는 대조 생쥐와 비교하여, 익숙한 물체에 비하여 새로운 물체의 향상된 식별을 보여주었다. 유사하게, 벡터 주사에 앞서, 대조 생쥐 및 항-Grik2 서열로 추후 처리되는 생쥐는 익숙한 물체 및 새로운 물체를 탐색하는 동안 유사한 거리를 이동하였다. 바이러스 벡터 주사 다음에, 항-Grik2 작제물 (G9; 서열 번호: 68)로 처리된 생쥐는 대조 생쥐 (다시 말하면, GC; 서열 번호: 101을 인코딩하는 바이러스 벡터가 주사된 생쥐)와 비교하여 감소된 이동 시간을 보여주었는데, 이것은 운동 탐색에 관여하는 감소된 성향 및 새로운 물체를 탐색하는 증가된 성향을 표시한다 (도 4c). NOR 과제 데이터의 정량 분석은 아래의 표 16 및 표 17에서 제공된다.
표 16: NOR 과제 데이터의 정량적 요약
표 17: 표 16에서 데이터의 유의성 분석 (p-값; ANOVA)
자연발생적 전자사진술 발작이, 해마내 EEG 전극이 이식된 생쥐로부터 차후에 기록되었다. 전자사진술 발작의 예시적인 전압 트레이스는 도 4d에서 제공된다. Grik2 표적화 ASO 작용제 (G9; 서열 번호: 68)를 인코딩하는 AAV 벡터로 처리된 생쥐는 스크램블 서열 (GC; 서열 번호: 101)을 제공받는 대조 생쥐에 비하여 5 일에 걸쳐, 누적 발작 지속 기간 (도 4e) 및 발작의 누적 횟수 (도 4f)에서 유의미한 감소를 나타냈다. 전술된 실험의 정량적 요약은 아래의 표 18에서 제공된다.
표 18: EEG 데이터의 정량적 요약
종합하면, 이들 조사 결과는 바이러스로 인코딩된, Grik2 표적화를 이용한 Grik2 mRNA의 발현의 저해가 TLE의 뮤린 모형에서 발작 빈도 및 지속 기간을 감소시키는 데 효과적이라는 것을 암시한다. 이런 이유로, 저해성 Grik2 표적화 ASO 작용제는 인간 환자에서 TLE의 치료를 위한 유망한 치료적 도정이다.
실시예 4:
Grik2
mRNA를 표적으로 하는 안티센스 올리고뉴클레오티드를 인코딩하는 바이러스 벡터의 투여에 의한, 인간 개체에서 뇌전증의 치료
뇌전증 (예를 들면, TLE, 예컨대 예를 들면, mTLE 또는 lTLE)을 겪는 것으로 진단된 개체, 예컨대 인간 개체 (예를 들면, 소아 또는 성인 개체)는 (a) 발작 재발의 위험; (b) CNS에서 흥분독성 및 연관된 뉴런 세포 사멸의 감소; (c) CNS의 영향을 받은 영역에서 생리학적 흥분 억제 균형의 복구; (d) 뇌전증의 1가지 이상의 증상 (예를 들면, 뇌전증 발작의 빈도, 지속 기간 또는 강도, 허약, 부재, 갑작스러운 착란, 언어를 이해하거나 생성하는 데 문제가 있음, 인지 장애, 손상된 가동성, 현기증, 또는 균형 또는 조화의 상실, 마비, 그리고 감정적 조절장애)에서 감소, 그리고 (e) 해마에서 치상회 과립 세포의 재발성 이끼 섬유의 병리학적 발아 중에서 1가지 이상 (예를 들면, 2가지 이상, 3가지 이상, 4가지 이상)을 포함하지만 이들에 한정되지 않는, 1가지 이상의 뇌전증 증상을 감소시키기 위해, 본원에서 설명된 조성물로 치료를 받을 수 있다. 치료 방법은 임의적으로, 환자를 진단하거나 또는 환자를 개체에서 본원 발명의 조성물을 이용한 치료를 위한 후보로서 확인하는 단계를 포함할 수 있다. 조성물은 Grik2 mRNA를 표적으로 하는 ASO 작용제, 또는 이것을 인코딩하는 폴리뉴클레오티드를 내포하는 핵산 벡터 (예를 들면, 바이러스 벡터, 예컨대 AAV 벡터, 예를 들면, AAV1, AAV2, AAV3, AAV4, AAV5, AAV6, AAV7, AAV8, AAV9, AAV10, AAV11, AAV12, AAV13, AAV14, AAV15, AAV16, AAV.rh8, AAV.rh10, AAV.rh20, AAV.rh39, AAV.Rh74, AAV.RHM4-1, AAV.hu37, AAV.Anc80, AAV.Anc80L65, AAV.7m8, AAV.PHP.B, AAV.PHP.EB, AAV2.5, AAV2tYF, AAV3B, AAV.LK03, AAV.HSC1, AAV.HSC2, AAV.HSC3, AAV.HSC4, AAV.HSC5, AAV.HSC6, AAV.HSC7, AAV.HSC8, AAV.HSC9, AAV.HSC10, AAV.HSC11, AAV.HSC12, AAV.HSC13, AAV.HSC14, AAV.HSC15 또는 AAV.HSC16에서 선택되는 혈청형 중에서 한 가지를 갖는 AAV 벡터, 또는 렌티바이러스 벡터)를 포함할 수 있다. 예시적인 ASO는 서열 번호: 1-100의 핵산 서열 중에서 하나에 무려 85% (예를 들면, 적어도 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 그 초과)의 서열 동일성을 가질 수 있거나, 또는 이들은 서열 번호: 1-100 중에서 하나 이상의 서열을 가질 수 있다. 게다가, 바이러스 벡터 (예를 들면, AAV 벡터)는 U.S. 특허가출원 번호: 63/050,742로부터 참조로서 편입되는, 표 9 또는 표 10에서 제공된 목록에서 선택되는 발현 카세트, 또는 도 30 또는 표 9에서 설명된 작제물 중에서 하나를 내포할 수 있다.
개체는 예를 들면, 정맥내, 복막내, 피하 또는 경피 투여를 비롯한, 임의의 적절한 수단에 의해, 또는 동물의 중추신경계에 직접적으로 투여 (예를 들면, 정위, 뇌실질내, 척수강내, 또는 뇌실내 주사)에 의하여 조성물이 투여될 수 있다. 조성물은 치료 효과량으로, 예컨대 개체마다 105, 106, 107, 108, 109, 1010, 1011, 1012, 1013, 1014 또는 1015개 유전체 사본 (GC)의 용량으로, 105, 106, 107, 108, 109, 1010, 1011, 1012, 1013 또는 1014개 GC/kg (개체의 전체 체중)의 용량으로, 환자의 뇌 질량의 그램당 105, 106, 107, 108, 109, 1010, 1011, 1012, 1013, 1014 또는 1015개 GC의 용량으로 투여될 수 있다. 개체의 뇌 중량 추정치는 MRI 뇌 용적 결정으로부터 획득되는데, 이것은 뇌 질량으로 전환되고 투여된 약물의 정확한 용량을 계산하는 데 활용된다. 뇌 중량은 또한, 공개된 데이터베이스를 이용하여, 연령 범위에 근거하여 추정될 수 있다. 작용제는 격월, 월 1회, 2 주마다 1회, 또는 적어도 주 1회 또는 그 초과 (예를 들면, 주 1, 2, 3, 4, 5, 6 또는 7회, 또는 그 초과)로 투여될 수 있다. 조성물은 두 번째 치료 양상, 예컨대 두 번째 치료제 (예를 들면, 항뇌전증 약물), 외과적 개입 (예를 들면, 외과적 적출, 방사선수술, 감마 나이프, 또는 레이저 절제), 미주 신경 자극, 뇌심부 자극, 또는 머리뼈경유 자성 자극과 병용 투여될 수 있다.
조성물은 (a) 발작 재발의 위험; (b) CNS에서 흥분독성 및 연관된 뉴런 세포 사멸의 감소; (c) CNS의 영향을 받은 영역에서 생리학적 흥분 억제 균형의 복구; (d) 뇌전증의 1가지 이상의 증상 (예를 들면, 뇌전증 발작의 빈도, 지속 기간 또는 강도, 허약, 부재, 갑작스러운 착란, 언어를 이해하거나 생성하는 데 문제가 있음, 인지 장애, 손상된 가동성, 현기증, 또는 균형 또는 조화의 상실, 마비, 그리고 감정적 조절장애)에서 감소, 그리고 (e) 해마에서 치상회 과립 세포의 재발성 이끼 섬유의 병리학적 발아 중에서 1가지 이상 (예를 들면, 2가지 이상, 3가지 이상, 4가지 이상)을 10% 또는 그 초과 (예를 들면, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95% 또는 그 초과)로 감소시키는 데 충분한 양으로 개체에게 투여될 수 있다. 뇌전증의 상기 열거된 증상은 표준 방법, 예컨대 신경학적 검사, 뇌파도, 뇌자도, CT 스캔, PET 스캔, fMRI 스캔, 비디오카메라촬영, 그리고 육안 관찰을 이용하여 사정될 수 있다. 치료의 효능을 평가하기 위해, 조성물의 투여 전후에 뇌전증 증상의 척도가 비교될 수 있다. 전술된 뇌전증의 증상에서 감소의 발견은 조성물이 개체에서 뇌전증을 성공적으로 치료했다는 것을 암시한다.
실시예 5: 마이크로RNA 골격을 이용하여,
Grik2
mRNA를 표적으로 하는 안티센스 발현 작제물의 설계
실시예 1A 및 실시예 1B에서 설명된 siRNA 스크린으로부터 전술된 선택 기준에 근거하여, 다른 설명된 가이드를 A-miR-30 골격 (S1) 내로 통합하는 추가의 작제물이 설계되었다 (도 6a-6e). 성숙 miRNA 발현과 처리를 향상시키기 위해, 이들 추가의 가이드 서열의 부분집합이 추가의 miRNA "골격" (내인성 miRNA으로부터 5' 측부 영역, 루프 서열 및 3' 측부 영역을 내포) 내로 통합되었다 (S2 = E-miR-30 골격; S3 = E-miR-155 골격; S4 = E-miR-218-1 골격; 및 S5 = E-miR-124-3 골격). hSyn 프로모터 (서열 번호: 790)의 제어하에, miRNA 발현 플라스미드의 이러한 더 큰 패널이 유도 만능 줄기 세포 (iPSC) 유래된 글루타민산성 뉴런 (GlutaNeurons) 내로 형질감염되고 Grik2 mRNA 수준을 감소시키는 능력에 대해 qPCR에 의해 평가되었다. 비형질감염된 세포 (파선)와 비교하고, 그리고 2의 중위 절대 편차 (MAD)를 이용하여 기능적 작제물 (점선)을 확인할 때, 대다수의 작제물은 기능적인 것으로 결정되었다 (다시 말하면, 이들은 MAD 미만에서 녹다운 Grik2 mRNA를 나타낸다)(도 5). 검사된 안티센스 서열은 G9 (서열 번호: 68), GI (서열 번호: 77), MW (서열 번호: 80), GU (서열 번호: 96), TO (서열 번호: 14), TK (서열 번호: 74), TH (서열 번호: 22), CQ (서열 번호: 35), XU (서열 번호: 51), XY (서열 번호: 83), Y9 (서열 번호: 88), YA (서열 번호: 63), GG (서열 번호: 91), G8 (서열 번호: 92), ME (서열 번호: 69), 그리고 MD (서열 번호: 70)를 포함하였다. 이들 중에서, GI (서열 번호: 77)-S2 (서열 번호: 798), MW (서열 번호: 80)-S4 (서열 번호: 799), MW-S5 (서열 번호: 800), 그리고 G9 (서열 번호: 68)-S5 (서열 번호: 801)가 Grik2 mRNA를 가장 높은 정도로 녹다운시키는 것으로 밝혀졌고, 그리고 이런 이유로 가장 기능적인 작제물 (다시 말하면, 20% 이상의 녹다운)로서 확인되었다.
단일 miRNA를 인코딩하는 플라스미드를 이용하여 제조된 AAV 벡터는 하기 이유로, 부적절하게 포장된 AAV를 야기할 수 있다. 첫째, 일차-miRNA 서열이 짧고 (<200개 염기), 그리고 프로모터 길이에 따라서, 단일 miRNA의 발현을 제어하는 단일 프로모터를 갖는 도입유전자 카세트의 설계가 AAV의 최대 포장 용량 (~4.8 kb)보다 훨씬 짧은 AAV 유전체를 야기할 수 있다. 이런 이유로, 만약 기대되는 전체 유전체 길이가 <2.4 kb (AAV의 포장 용량의 절반)이면, 단일 캡시드에 하나 초과의 벡터를 부하하는 것이 가능할 수 있다. 이것은, 만약 적합한 길이이면 AAV 캡시드 내로 포장될 수 있는 AAV 유전체 이합체 (또는 삼합체)의 생산을 가능하게 하는 적절한 엔도뉴클레아제 닉킹 없이, 중합효소 번역 초과에 의해 매개될 수 있다. 이것은 차후에, AAV 벡터 입자의 모집단 내로 유의미한 이질성을 도입하는데, 이로 인해 의약품의 제조와 특징화가 훨씬 어려워진다.
둘째, shRNA- 및 miRNA 기반 도입유전자가 miRNA 헤어핀의 포함으로 인해, 생래적으로 유의미한 이차 구조를 갖는다. AAV 유전체 내에 이들 내부 이차 구조는 AAV 유전체 복제와 포장 동안 "가성" ITR로서 기능하여, 바람직하지 않은 절두 사건, 그리고 완전한 및 부분적인 벡터의 혼합물을 내포하는 AAV 벡터 입자의 이질성 모집단을 야기할 수 있는 것으로 밝혀졌다.
우리는 적절하게 포장된 AAV를 생산하는 능력에 대해, 여러 작제물을 검사하였다. 이들 작제물의 설계에서 실행된 이들 전략은 전술된 부적절한 AAV 유전체 포장의 과제를 극복한다.
다수의 상이한 길이와 형식의 합성 miRNA를 인코딩하는 AAV 벡터가 생성되었고 (도 6a-6e), 유전체가 추출되고 유전체 길이와 무결성을 평가하기 위해 알칼리성 겔 전기이동에 의해 평가되었다 (도 7). 단일 프로모터 및 단일 miRNA 카세트 (예상된 길이: 1.5 kb)를 인코딩하는 플라스미드로부터 생성된 벡터 프렙의 유전체 내용물은 단독적으로 (1.5 kb), 이중으로 (3.0 kb) 및 삼중으로 (4.5 kb) 포장된 유전체의 혼합물로 구성되는 것으로 밝혀졌는데, 이것은 짧은 길이의 프로모터-miRNA 도입유전자 카세트가 단량체성, 이합체성 및 삼합체성 유전체의 포장을 허용하고 벡터 중 단지 57%만 원하는 단량체이라는 것을 표시한다 (농도측정에 의해, 표 19).
표 19: 유전체 길이의 함수로서 단독적으로 포장된 벡터 유전체의 백분율
두 번째 프로모터가 첫 번째 프로모터의 상류에 추가될 때 (탠덤 프로모터를 야기하고 전장 유전체 길이를 AAV의 포장 용량의 절반보다 약간 더 많은 2.9kb로 확장), 거의 모든 벡터 프렙 유전체가 전장 (97%)이다. miRNA 헤어핀의 포함은 이들 2가지 유전체 형식 중 어느 쪽에서도 절두 사건을 도입하지 않았는데, 그 이유는 포장된 유전체 길이 모두 단량체성, 전장 형태의 배수이었기 때문이다.
효과적인 유전체 길이를 증가시키는 한 가지 방법은 벡터의 효과적인 길이를 배증하는, 이중 가닥, 자가 상보성 유전체를 갖는 AAV를 생산하는 것이다. 이것은 돌연변이체 ITR (mITR), 이 경우에 있어서 5' ITR을 도입함으로써 달성된다. 2개의 scAAV 벡터 (각각, 단일 프로모터 및 단일 miRNA를 포함): 야생형 (wt) ITR에 인접한 프로모터를 포함하는 것 및 mITR에 인접한 프로모터를 포함하는 것이 유전체 무결성에 대해 평가되었다. 각 배향에 대한 대다수의 벡터는 전장 (각각, 2.6kb 65 및 56%)인 것으로 밝혀졌다; 하지만, 더 작은 분자량 유전체 단편뿐만 아니라 아마도 부분적으로 이합체성 유전체 (예를 들면, 하나의 전장 유전체 + 하나의 부분적인 절두된 단편)로 구성되는 더 큰 분자량 유전체를 야기하는 유의미한 유전체 절두 사건의 증거가 있었다. ~5.2 kb에서 덜 두드러진 종 역시, 전장 이합체성 scAAV 유전체가 또한 존재한다는 명백한 증거이었다.
miRNA 발현을 또한 증가시키면서 유전체 길이를 증가시키는 다른 방법은 추가의 miRNA 모티프 (단일 miRNA의 복수 사본, 복수의 miRNA의 단일 사본, 또는 이들의 조합)의 도입이다; 별개의 성숙, 기능적 miRNA를 생성하는, 복수의 miRNA의 발현 또한, 소정의 mRNA의 원위 영역을 표적화하는 잠재력을 도입한다. 2.2kb의 예상된 전장 유전체를 각각 야기하는, 연쇄체화된 miRNA를 포함하는 2개의 작제물: G9 (서열 번호: 68)-S1의 3개 사본을 포함하는 작제물 및 G9-S1, GI (서열 번호: 77)-S1 및 MU (서열 번호: 96)-S1 각각의 1개 사본을 포함하는 작제물이 검사되었다 (도 7). 이들 유전체가 AAV의 포장 용량의 절반보다 크지 않기 때문에, 이중 포장의 증거가 있었고 유전체 모집단 중 단지 30%만 원하는, 단량체성 전장 유전체이었다. 추가적으로, 다수의 부분적인 유전체가 검출되었는데, 이들의 길이는 전체 유전체 모집단 내로 유의미한 이질성을 도입하는, 3개의 카세트 모두에 대한 S1 골격의 포함으로 인한 개별 miRNA 카세트 측접 서열 내에 상동성 서열 사이의 재조합 사건을 암시하였다. 이들 조사 결과는 벡터 길이를 최대 벡터 용량에 더 가깝게 가져가기 위한, 추가 서열의 단일-miRNA 발현 작제물 내로의 도입이 결과적인 유전체 이질성을 감소시키고 전장 벡터를 더 신뢰성 있게 생성한다는 것을 암시한다.
실시예 6:
Grik2
mRNA를 표적으로 하는 이중-안티센스 발현 작제물은 AAV9 벡터에서 단독적으로 포장된다
실시예 5에서 상기 조사 결과에 근거하여, 이러한 세팅에서 AAV 유전체 모집단의 균질성이 ssAAV 형식의 길이뿐만 아니라 이용에 의해 주동된다. 유사하게 염기서열분석된 miRNA를 연쇄체화하는 함정을 방지하면서, 이들 요건에 부합하는 작제물을 구축하기 위해, 2개의 프로모터, 2개의 miRNA 카세트 전략이 채택되었다. 이러한 형식에서, 하나의 프로모터 (여기에서 hSyn (서열 번호: 790)으로서 예시됨)가 하나의 폴리A 서열에서 종결되는, 단일 miRNA의 발현을 하나의 골격에서 주동하고, 이후에 뒤따르는 두 번째 프로모터 (여기에서 CaMKII로서 예시됨)가 두 번째 폴리A 서열에서 종결되는, 두 번째 miRNA의 발현을 상이한 골격에서 주동한다.
이러한 접근방식은 유전체 내에 서열 상동성을 최소화하고, 그리고 이합체성 또는 삼합체성 유전체를 포장하는 것을 방지할 만큼 충분히 긴 (~3.7 kb, 단독적으로 포장된, 전장 AAV에 대한 길이 요건 내에) 유전체를 야기한다. 도 7에서 도시된 플라스미드 스크린의 결과에 근거하여, 상위 "히트" (DMTPV1-4)의 고유한 miRNA-골격의 조합을 이용하여 4개의 이런 "이중 작제물"이 만들어졌다.
DMTPV1은 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 (서열 번호: 790), E-miR-124-3 5' 측부 서열 (서열 번호: 768), G9의 안티센스 서열 (서열 번호: 68)에 상보적인 센스 승객 가닥 서열, E-miR-124-3 루프 서열 (서열 번호: 770), G9의 안티센스 가이드 서열, E-miR-124-3 3' 측부 서열 (서열 번호: 769), 소 성장 호르몬 (BGH) 폴리A 서열 (서열 번호: 793), CaMKII 프로모터 서열 (서열 번호: 802), E-miR-30 5' 측부 서열 (서열 번호: 759), GI (서열 번호: 77)에 상보적인 센스 승객 가닥 서열, E-miR-30 루프 서열 (서열 번호: 761), GI의 안티센스 가이드 서열 (서열 번호: 77), E-miR-30 3' 측부 서열 (서열 번호: 760), 토끼 베타-글로빈 (RBG) 폴리A 서열 (서열 번호: 792), 그리고 3' ITR 서열 (서열 번호: 748)(도 8a)을 내포하였다.
DMTPV2는 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 (서열 번호: 790), E-miR-124-3 5' 측부 서열 (서열 번호: 768), G9의 안티센스 서열 (서열 번호: 68)에 상보적인 센스 승객 가닥 서열, E-miR-124-3 루프 서열 (서열 번호: 770), G9의 안티센스 가이드 서열, E-miR-124-3 3' 측부 서열 (서열 번호: 769), BGH 폴리A 서열 (서열 번호: 793), CaMKII 프로모터 서열 (서열 번호: 802), E-miR-218 5' 측부 서열 (서열 번호: 765), MW (서열 번호: 80)에 상보적인 센스 승객 가닥 서열, E-miR-218 루프 서열 (서열 번호: 767), MW의 안티센스 가이드 서열 (서열 번호: 80), E-miR-218 3' 측부 서열 (서열 번호: 766), RBG 폴리A 서열 (서열 번호: 792), 그리고 3' ITR 서열 (서열 번호: 748)(도 8b)을 내포하였다.
DMTPV3은 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 (서열 번호: 790), E-miR-30 5' 측부 서열 (서열 번호: 759), GI (서열 번호: 77)에 상보적인 센스 승객 가닥 서열, E-miR-30 루프 서열 (서열 번호: 761), GI의 안티센스 가이드 서열 (서열 번호: 77), E-miR-30 3' 측부 서열 (서열 번호: 760), BGH 폴리A 서열 (서열 번호: 793), CaMKII 프로모터 서열 (서열 번호: 802), E-miR-124-3 5' 측부 서열 (서열 번호: 768), G9의 안티센스 서열 (서열 번호: 68)에 상보적인 센스 승객 가닥 서열, E-miR-124-3 루프 서열 (서열 번호: 770), G9의 안티센스 가이드 서열, E-miR-124-3 3' 측부 서열 (서열 번호: 769), RBG 폴리A 서열 (서열 번호: 792), 그리고 3' ITR 서열 (서열 번호: 748) (도 8c)을 내포하였다.
DMTPV4는 5'에서 3'로, 5' ITR 서열 (서열 번호: 746), hSyn 프로모터 (서열 번호: 790), E-miR-30 5' 측부 서열 (서열 번호: 759), GI (서열 번호: 77)에 상보적인 센스 승객 가닥 서열, E-miR-30 루프 서열 (서열 번호: 761), GI의 안티센스 가이드 서열 (서열 번호: 77), E-miR-30 3' 측부 서열 (서열 번호: 760), BGH 폴리A 서열 (서열 번호: 793), CaMKII 프로모터 서열 (서열 번호: 802), E-miR-124-3 5' 측부 서열 (서열 번호: 768), MW의 안티센스 서열 (서열 번호: 80)에 상보적인 센스 승객 가닥 서열, E-miR-124-3 루프 서열 (서열 번호: 770), MW의 안티센스 가이드 서열 (서열 번호: 80), E-miR-124-3 3' 측부 서열 (서열 번호: 769), RBG 폴리A 서열 (서열 번호: 792), 그리고 3' ITR 서열 (서열 번호: 748) (도 8d)을 내포하였다.
이들 이중 작제물은 AAV9 벡터로서 생산되었고, 그리고 유전체 무결성에 대한 2개의 프로모터, 2개의 miRNA 카세트 전략의 영향이 평가되었다. 2개의 단일 프로모터, 단일 miRNA 벡터 (전장 = 1.5 kb, 이합체성과 삼합체성 포장의 증거를 나타냄)와 비교할 때, 이들 이중 작제물 벡터의 유전체 내용물은 ~3.7 kb의 예상된 전장 크기에서 단일 띠의 존재에 의해 증거된 바와 같이, 균일한 것으로 밝혀진다 (도 9a 및 도 9b). 이런 이유로, 두 번째 프로모터 및 두 번째 miRNA 카세트의 도입은 표적화 효율 및 전체 miRNA 발현을 향상시키기 위한 목적으로, 유전체 무결성을 유지하고, 절두 사건을 방지하며, 복수의 miRNA의 발현을 허용하면서 유전체가 단량체로서 포장되도록, AAV 유전체의 길이를 증가시키는 효과적인 방법이다.
실시예 7: 2개의 구별되는 항-
Grik2
miRNA 서열의 조합을 이용한
Grik2
mRNA 녹다운의 시험관내 효능
우리는 먼저, 2가지 상이한 miRNA 가이드가 동일한 벡터 시스템에서 또는 2개의 별개의 벡터로서 발현될 때, Grik2 발현의 동등하진 않지만 효율적인 녹다운을 매개할 수 있는지를 검사하였다. 이중 작제물 벡터 내로 조합되는 4개의 플라스미드 작제물이 선택되었다 (도 7에서 조사 결과에 근거하여). 이중 작제물 벡터의 miRNA 구성요소가 단독적으로 또는 서로 조합으로 GlutaNeurons에 형질감염되었고 (형질감염된 플라스미드의 총량을 일정하게 유지), 그리고 Grik2 mRNA 수준이 qPCR에 의해 계측되었다 (도 10). 함께 형질감염될 때, 항-Grik2 miRNA의 조합 (둘 모두 시냅신 프로모터의 제어하에)은 견실한 Grik2 mRNA 녹다운을 매개할 수 있었는데, 이것은 Grik2에 대하여 하나 초과의 고유한 가이드를 발현하는 벡터의 이용을 뒷받침하였다.
우리는 이후, 뇌전증모양 활성을 억제하는 능력에 대해 단일 프로모터 (hSyn)를 포함하는 상이한, 단일 miRNA 카세트를 내포하는 항-Grik2 miRNA-인코딩 벡터 제조물의 능력을 아래와 같이 검사하였다:
생쥐 해마 기관형적 슬라이스:
기관형적 슬라이스가 기존 문헌 (Peret et al. 2014)에서 설명된 바와 같이, McIlwain 조직 세단기를 이용하여 WT Swiss 생쥐 (P9-P10)로부터 준비되었다. 슬라이스가 1 mL의 하기 배지: MEM 50%, HS 25%, HBSS 25%, HEPES 15 mM, 글루코오스 6.5 mg/mL 및 인슐린 0.1 mg/mL를 내포하는 배양 접시 내부에 그물망 삽입물 (Millipore) 상에 배치되었다. 배양 배지가 2-3 일마다 교체되었고, 그리고 슬라이스가 37 ℃/5% CO2에서 인큐베이터 내에 유지되었다. 필로카르핀 (0.5 μM)이 5 DIV에 배지에 첨가되고 7 DIV에 제거되었다; 슬라이스가 9 DIV부터 11 DIV까지 실험에 활용되었다.
기관형적 슬라이스의 바이러스 감염
생쥐 기관형적 슬라이스의 감염을 위해, AAV9 작제물을 내포하는 1μL의 배지 (인산염 완충된 식염수)가 DIV0에 슬라이스 상에 직접적으로 떨어뜨려졌다.
전기생리학적 기록
생쥐 기관형적 슬라이스가 30-32℃에 유지된 기록 챔버로 개별적으로 이전되고, 그리고 5 μM 가바진 (Sigma-Aldrich)의 존재에서 산소화된 (95% O2 및 5% CO2) ACSF; NaCl, 3.5 KCl, 1.2 NaH2PO4, 26 NaHCO3, 1.3 MgCl2, 2.0 CaCl2 및 10 글루코오스 (pH ~7.4)(Sigma-Aldrich) (mM로)를 내포하는 ACSF로 연속적으로 관류되었다 (2-3 mL/분). 지역장 전위 (LFP) 기록이 치상회의 과립 세포 층에 배치된 단극 니크롬선으로 이루어졌다. DAM-80 증폭기가 기록에 이용되었다 (저역 필터, 0.1 Hz; 고역 필터, 3 KHz; World Precision Instruments, Sarasota, FL); 데이터가 Digidata 1440A (Molecular Devices)를 이용하여 컴퓨터로 디지털화되고 (20 kHz), 그리고 Clampex 10.1 소프트웨어 (PClamp, Molecular Devices)를 이용하여 획득되었다. 신호가 Clampfit 9.2 (PClamp) 및 MiniAnalysis 6.0.1 (Synaptosoft, Decatur, GA)을 이용하여 오프라인 분석되었다.
통계
모든 값은 평균 ± SEM으로서 제공된다. 통계학적 분석이 GraphPad Prism (GraphPad software 5.01)을 이용하여 수행되었다.
검사 작제물: G9, GI, G9+GI
검사된 벡터, G9 및 GI (아래의 표 20 참조)는 hSyn 프로모터 하에 AAV9 바이러스 벡터에 의해 전달된 miRNA 서열에 상응한다. 대조 벡터, GC는 hSyn 프로모터의 제어하에 AAV9 바이러스 벡터에 의해 전달된 GFP 및 스크램블 miRNA 서열 (GC, 서열 번호: 101)을 포함하였다.
표 20: 슬라이스 전기생리학 실험에 대한 벡터 상세
G9- 및 GI-인코딩 작제물은 혼합되고 (단독적으로 전달된 것들과 동등한 부분, 동일한 총 벡터 사본), 그리고 생쥐 기관형적 슬라이스에 적용될 때, 뇌전증모양 활성을 각 벡터 프렙과 적어도 동등한 정도로 개별적으로 감소시키는 것으로 밝혀졌다 (도 11 및 아래의 표 21과 22).
표 21: 슬라이스 전기생리학 데이터 요약
표 22: ANOVA를 이용한 표 21에서 데이터의 통계학적 분석
이후, Grik2 mRNA 녹다운을 매개하는 이중-miRNA AAV9 벡터의 능력이 인간 iPSC 유래된 GlutaNeurons에서 평가되었다. 세포가 PEI 및 라미닌으로 피복된 384 웰 평판에서 17,500개 세포/웰의 밀도로 파종되고, 11 일 동안 배양되고, 3x105개 GC/세포의 MOI로 AAV 벡터로 형질도입되고, 그리고 형질도입후 8 일째에 qPCR 분석을 수확되었다. AAV9.null 벡터 (RNA를 생산하지 않는 전장 유전체를 내포하는 AAV9, 검은색 막대)로 형질도입된 세포와 비교하고, 그리고 2의 중위 절대 편차 (MAD)를 이용하여 기능적 작제물 (점선, 값 = 0.9)을 확인할 때, 이중 AAV9 작제물 (체크 막대)은 이러한 시점에서 이러한 MOI에서 그들의 단일 작제물 구성요소 부분 (hSyn 구성요소 = 회색 막대, CaMKII 구성요소 = 흰색 막대)보다 더 높은 정도는 아니지만, 적어도 동등한 정도로 유의미한 Grik2 mRNA 녹다운을 매개하는 것으로 결정되었다 (도 12).
실시예 8: 측두엽 뇌전증의 필로카르핀 유발 뮤린 모형에서
Grik2
mRNA 표적화 단일- 및 이중-miRNA AAV9 발현 작제물의 생체내 효능
우리는 TLE의 뮤린 필로카르핀 유발 모형을 이용하여, 뇌전증 지속증의 유도 이후에 다양한 발현 작제물의 투여와 함께 생체내에서 Grik2 mRNA 표적화 이중-miRNA AAV 발현 벡터의 효능을 사정하였다.
재료 & 방법
윤리학
모든 실험은 유럽 의회 지침 (2010/63/UE)에 따라서, 지역 윤리 위원회 (동의 번호 APAFIS#9896-201605301121497v11)에 의한 평가 이후에, Institut National de la Sant et de la Recherche Mdicale (INSERM) 동물실험윤리위원회에 의해 승인되고 Ministre de l'Education Nationale, de l'Enseignement Suprieur et de la Recherche에 의해 공인되었다.
만성 뇌전증 생쥐
실험이 수컷 Swiss 생쥐를 이용하여 수행되었다. 이들 생쥐는 실온 (20-22℃)에서 12 시간 명암 주기에 유지되었고 사료와 물에 무제한으로 접근 가능하였다. 생쥐 (30-40 g)는 필로카르핀 (300-350 mg/kg)의 복막내 투여 30 분 전에 스코폴라민 (1 mg/kg)이 피하 (s.c.)로 제공되었다. 램프 프로토콜이 이용되었는데, 여기서 동물은 300 mg/kg의 초기 용량, 그 이후에 발작이 나타날 때까지 30 분마다 절반 용량이 제공되었다. WT 생쥐는 전형적으로, 뇌전증 지속증 (SE)이 시작되기 전 적어도 2번의 발작을 경험하였다. 첫 번째 발작 10 분 후에 카페인 (40 mg/kg)이 투여되었다. 뇌전증 지속증 (SE)의 개시 후 1 시간째에 디아제팜 (10 mg/kg)이 투여되었다.
AAV 주사
뇌전증 생쥐 (SE 후 >2 개월)가 이소플루란 마취하에 마취되었다 (100% O2 하에 유도의 경우 5% 및 유지의 경우 2%). 체온이 보온 패드를 이용하여 유지되었고, 그리고 이들은 정위 프레임에 배치되었다. 리도카인 (2%)의 국부 투여의 존재에서, AAV를 해마의 등면 및 복면 치상회 내로 양측으로 주사하기 위해 4개의 홀이 천공되었다 (AP -1.8 mm, ML ±1 mm, DV - 2 mm, 그리고 AP -3.3 mm, ML ± 2.3 mm, DV - 2.5 mm). 각 주사를 위해, Hamilton 주입기가 이용되었다. 뇌 조직이 삽관 위를 미끄러져 가도록 하기 위한 5-분 지연 후, AAV를 내포하는 용액의 용적 = 1.0 μL/주사 부위 x 4 주사 부위가 천천히 주입되었다 (속도: 0.2 μL/분). 주입 후, 주입기 트랙을 따라서 용액의 역류를 예방하기 위해, 주입기가 5 분 더 제자리에 남겨졌다. 회복 동안, 이들 동물은 24 및 48 시간 후 5 mg/kg s.c. 카르프로펜 (Rimadyl®)이 제공되었다.
운동 활성
비-뇌전증 및 뇌전증 생쥐 (SE 후 >2 개월)의 운동이 AAV 주사 1 주 전에 및 2 주 후에 평가되었다. 생쥐가 환경에 익숙해지도록, 실험 하루 전에 행동 분석실로 이전되었다; 이들 생쥐는 실온 (20-22℃)에서 9:00-18:00 명암 주기로 유지되었고 사료와 물에 무제한 접근이 가능하였다. 검사된 동물과 접촉된 모든 재료는 그 후에, 후각 신호를 예방하기 위해 아세트산으로 세척되었다. 먼저, 자발적 탐색 행동이 오픈 필드 시험으로 검사되었다. 간단히 말하면, 생쥐가 10 분 동안 50 x 50 x 50 cm 청색 폴리염화비닐 상자의 중심에 배치되었고, 그리고 궤적이 추적 소프트웨어 EthoVision Color (Noldus, The Netherlands)에 연결된 비디오 카메라로 기록되었다; 5-분 탐색 동안 생쥐에 의해 커버된 속도 및 총 거리가 분석되었다.
EEG
AAV 주사 3 주 후에 하나의 뎁스 와이어 전극이 뇌전증 생쥐 (SE 후 >2 개월)에게 이식되었다; 전술된 바와 같이, 수술이 이소플루란 마취하에 수행되었다. 전극이 치상회 (DG) 내로 주촉성으로 배치되었다 (정수리점으로부터 Paxinos 및 Watson 좌표: AP -2.55 mm, ML +1.65 mm, DV -2.25 mm). 소뇌 위에 배치된 추가 스크류가 접지 전극으로서 역할을 하였다. 상기 전극 및 스크류는 치과용 시멘트로 두개골 위에 고정되었다. 회복 동안, 이들 동물은 5 mg/kg s.c. 카르프로펜 (Rimadyl®)이 24 및 48 시간 후 제공되었다. EEG (증폭되고 (1000X), 0.16-97 Hz 통과에서 필터링되고, 500 Hz에서 획득됨)가 텔로미터 시스템을 이용하여 5 일 동안, 하루 24 시간 모니터링되었다 (Data Sciences International, St. Paul, MN). 해마내 EEG 트레이스는 DG 내로 삽입된 전극 및 소뇌 위에 배치된 전극 사이에 전위의 차이를 나타냈다.
통계
모든 값은 평균 ± SEM으로서 제공된다. 통계학적 분석이 Graphpad Prism 7 (GraphPad Software, La Jolla, CA)을 이용하여 수행되었다. 집단간 비교를 위해, 미가공 데이터가 만 휘트니 검정에 의해 분석되었다. 복수의 집단 비교를 위해, 미가공 데이터가 일원 변량 분석 검사에 의해 분석되었다. 유의성 수준은 p < 0.05. *p< 0.05, **p<0.01, ***p<0.001로 설정되었다.
결과
검사 항목
검사된 벡터, G9 (서열 번호: 68), GI (서열 번호: 77)는 hSyn 프로모터 (서열 번호: 790) 하에 AAV9 바이러스 벡터에 의해 전달된 miRNA 서열에 상응한다. GC (서열 번호: 101) 및 AAV9.GFP는 대조 벡터에 상응하고; GC 및 AAV9.GFP는 hSyn 프로모터 하에 AAV9 바이러스 벡터에 의해 전달된 GFP 및 스크램블 miRNA 서열, 그리고 GFP 서열을 각각 포함한다 (아래의 표 23 참조).
표 23: miRNA 단일 작제물에 대한 상세
G9 및 GI miRNA 작제물의 효능의 평가
G9 및 GI의 효능이 만성 뇌전증 생쥐 (뇌전증 지속증 이후에 >2 개월)에서 뇌전증의 행동 마커인 과운동, 그리고 자연발생적 재발성 발작이 평가되었다. 실험의 이러한 세트에서, GC가 대조 벡터로서 이용되었다.
과운동
G9- 및 GI-인코딩 벡터는 비-뇌전증 생쥐와 유사한 수준으로 향한, 과운동의 유의미한 감소를 나타냈다 (도 13a 및 도 13b 참조). 대조 벡터인 GC에서는 유의미한 변화가 관찰되지 않는다 (아래의 표 24 참조).
표 24: 오픈 필드 시험에서 이동된 총 거리
EEG
G9- 및 GI-인코딩 작제물은 대조 벡터인 GC-인코딩 작제물과 비교하여 뇌전증 발작의 억제를 나타냈다 (도 14 및 아래의 표 25 참조).
표 25: 하루에 발작 횟수
G9-인코딩 AAV9 작제물의 용량 반응
AAV9 발현 벡터에 인코딩된 G9를 이용한 용량 반응 검사가 만성 뇌전증 생쥐 (SE 이후에 >2 개월)에서 수행되었다. 상이한 희석하에 G9의 용량 반응의 효능 (아래의 표 26 참조)이 뇌전증의 행동 마커인 과운동에서, 그리고 자연발생적 재발성 발작에서 평가되었다. 실험의 이러한 세트에서, AAV9-GC가 대조 벡터로서 이용되었다.
표 26: 연구에 이용된 miRNA 작제물에 대한 상세
과운동
G9는 용량 반응 효능을 나타냈다; 우리는 주사 전후에 및 GC와 비교될 때 G9 및 G9/10의 유사한 효능, 그리고 G9/1000의 효능의 상실을 관찰하였다 (도 15 및 아래의 표 27 참조).
표 27: 이동된 총 거리
EEG
초기 데이터는 생쥐에서 G9-인코딩 작제물에 의한, 뇌전증 발작의 용량 의존성 억제를 암시한다 (도 16 및 아래의 표 28 참조).
표 28: 하루에 발작 횟수
이중-miRNA AAV9 발현 작제물
생체내에서 Grik2 mRNA 표적화 이중-miRNA AAV 발현 벡터의 효능을 결정하기 위해, TLE의 뮤린 필로카르핀 유발 모형이 뇌전증 지속증의 유도 이후에 다양한 단일-siRNA 및 이중-miRNA AAV 발현 작제물의 투여와 함께 이용되었다.
실험의 일반적인 구조는 다음과 같았다. 먼저, 뇌전증 지속증이 필로카르핀의 전신 투여를 통해 생쥐에서 유도되었고, 그리고 차후에 디아제팜으로 종결되었다. 생쥐는 회복할 수 있도록 약 1 주가 부여되었다. 그 다음 3 주에 걸쳐, 필로카르핀 처리된 생쥐는 그들이 뇌전증 발작의 안정된, 자연발생적 재발을 나타낼 때 (필로카르핀 처리의 시점으로부터 약 2 개월)까지 발작 발달에 대해 모니터링되었다. 일단 이러한 벤치마크가 도달되면, 생쥐는 재발성 뇌전증 발작의 확립 이후에 첫 번째 주 동안 오픈 필드에서 운동에 대해 검사되었다. 2-3 주 차 동안, 생쥐는 복수의 단일-siRNA 또는 이중-miRNA AAV 발현 벡터 중에서 하나로 처리되었다. 단일-siRNA 발현 벡터는 hSyn 프로모터 (서열 번호: 790)의 제어하에 스크램블 대조 siRNA 서열 (GC, 서열 번호: 101), 2개의 Grik2 표적화 서열 중에서 하나 (G9, 서열 번호: 68; 또는 GI, 서열 번호: 77), 또는 GFP 도입유전자를 포함하였다. 이중-miRNA 발현 작제물은 전술되고 아래의 표 29에 상세하게 나타나 있는 DSPTV1-4 작제물 중에서 하나를 포함하였다.
표 29: miRNA 이중 작제물에 대한 상세
처리후 약 15 일 후에, 오픈 필드 시험을 이용하여, 필로카르핀 및 여러 siRNA 발현 작제물 중에서 하나로 처리된 생쥐에서 다시 한 번 운동이 사정되었다. DSPTV3 및 DSPTV4 작제물은 생쥐에서 필로카르핀 유발 과운동의 유의미한 억제를 나타냈다 (도 17 및 아래의 표 30). AAV9-GFP가 대조 벡터로서 이용되었다.
표 30: 이동된 총 거리
발작 및 과운동 사이의 상관
생쥐의 오픈 필드 시험에서 과운동의 계측을 통해 Grik2 표적화, AAV-인코딩된 miRNA 작제물의 항-뇌전증유발 효과에 관한 결론을 도출하기 위해, 발작의 횟수 및 처리후 과운동 사이의 상관이 분석되었다. 발작의 극단값은 이상점 분석 ROUT (Q = 1.000%)에 근거하여 배제되었다. 전술된 실험 조건하에, 하루에 뇌전증 발작의 횟수 및 오픈 필드 시험에서 실험 생쥐에 의한 이동된 총 거리 사이에 유의미한 상관 (R2 = 0.7388, p < 0.0001)이 있었다 (도 18). 따라서, 과운동의 척도는 생쥐에서 뇌전증 상태에 대한 지수로서 이용되어, Grik2 표적화 안티센스 작제물의 항-뇌전증유발 효과를 검사하는 빠르고 인도적인 접근방식을 실시가능하게 할 수 있다.
결론
이들 데이터는 Grik2 mRNA를 표적으로 하는 RNAi 서열을 보유하는 AAV 벡터를 이용한 Grik2 유전자 침묵이 TLE에서 자연발생적인 만성 발작을 예방하는 데 효율적인 전략이라는 것을 증명한다.
실시예 9: 생쥐에서 이중 항-
Grik2
작제물을 인코딩하는 벡터의 시험관내와 생체내
효능
Grik2를 내생적으로 발현하는 세포에서 항-Grik2 서열을 내포하는 이중-miRNA 인코딩 벡터의 효능을 평가하도록 의도된 실험의 첫 번째 세트에서, GlutaNeurons가 단일 항-Grik2 miRNA 서열 G9 (서열 번호: 68) 또는 GI (서열 번호: 77)을 인코딩하는 AAV9 벡터, 이중 항-Grik2 서열 인코딩 벡터 DMTPV1 (서열 번호: 785), DMTPV2 (서열 번호: 786), DMTPV3 (서열 번호: 787) 또는 DMTPV4 (서열 번호: 788), 또는 지질 기반 형질감염 시약 내로 통합된 RNA-null 벡터로 형질도입되었다. TaqManTM 단일플렉스 실시간 정량적 중합효소 연쇄 반응 (RT-qPCR)이, 형질감염후 3개의 복제물을 이용하여 384-웰 평판 내에 GlutaNeurons에서 Grik2 녹다운의 수준을 정량하는 데 이용되었다. GlutaNeurons는 17,500개 세포/웰 (17.5k c/w)의 밀도로 도말되었다. Grik2 mRNA 발현이 GAPDH 신호에 대해 정규화되었다. Grik2 안티센스 작제물을 인코딩하는 AAV9 벡터의 형질도입은 G9, GI 및 DMTPV1-4를 내포하는 작제물의 경우에, 5 일 후 GlutaNeurons에서 Grik2 mRNA 발현을 유의미하게 감소시키는 것으로 밝혀졌다 (도 19). 이들 조사 결과는 Grik2 유전자를 내생적으로 발현하는 세포에서 Grik2 mRNA의 발현을 녹다운시키는, Grik2를 표적으로 하는 이중-miRNA 인코딩 벡터의 유용성을 증명한다.
실험의 두 번째 세트에서, G9 (서열 번호: 68), DMTPV3 (서열 번호: 787), AAV9.hSyn.GFP를 인코딩하는 벡터로 처리된 생쥐에서 오픈 필드 시험으로, 또는 처리되지 않은 생쥐에서 과거 대조로 자발적 탐색 행동이 검사되었다. 간단히 말하면, 생쥐가 10 분 동안 50 x 50 x 50 cm 청색 폴리염화비닐 상자의 중심에 배치되었고, 그리고 궤적이 추적 소프트웨어 EthoVision Color (Noldus, The Netherlands)에 연결된 비디오 카메라로 기록되었다; 10-분 탐색 동안 생쥐에 의해 커버된 총 거리가 분석되었다. G9 및 DMTPV3으로 처리된 생쥐는 과운동 활성에서 유의미한 감소 (p < 0.01)를 보여주었다 (도 20).
생쥐에서 과운동 활성에 대한 DMTPV3의 효과의 용량 의존성이 아래의 표 31에 나타나 있는 4가지 용량 전체에서 검사되었다.
표 31: 연구에 이용된 miRNA 작제물에 대한 상세
DMTPV3 및 DMTPV3/10 벡터는 생쥐에게 투여 이후에 과운동 활성의 유의미한 감소 (p < 0.05; 만 휘트니 검정; 도 21)를 생성하고, 그것에 의하여 생쥐에서 운동 활성에 대한 이중-안티센스 벡터의 용량 의존성 효과를 보여주었다.
Grik2 mRNA를 표적으로 하는 단일 및 이중 miRNA를 인코딩하는 벡터의 항-뇌전증유발 효과는 또한, 필로카르핀 처리된 생쥐에서 단일 G9 (서열 번호: 68) 및 GI (서열 번호: 77) 안티센스 서열을 인코딩하는 벡터, 이중-miRNA DMTPV3 (서열 번호: 787) 벡터, 또는 스크램블 서열 GC (서열 번호: 101) 또는 AAV9.hSyn.GFP를 인코딩하는 대조 벡터를 이용하여 검사되었다. G9, GI 및 DMTPV3으로 처리된 생쥐는 하루 동안 경험하는 발작의 횟수에서 유의미한 감소를 나타냈으며, DMTPV3이 G9 또는 GI 단독보다 훨씬 큰 감소를 나타냈다 (도 22). 이들 조사 결과는 Grik2 mRNA를 표적으로 하는 이중-miRNA 작제물이 생쥐에서 발작 활성을 억제하는 데 높은 효능을 보여준다는 것을 증명한다.
실시예 10: 인간 뇌전증 환자로부터 획득된 기관형적 해마 슬라이스에서
Grik2
mRNA 표적화 AAV-인코딩된 안티센스 작제물을 이용한 GluK2 단백질 발현의 녹다운
인간 뇌 조직에서 GluK2 단백질 발현을 녹다운시키는, Grik2 mRNA 표적화 안티센스 올리고뉴클레오티드를 인코딩하는 AAV9 발현 벡터의 효능을 사정하기 위해, 기관형적 해마 슬라이스가 7명의 인간 TLE 환자 (H7, H8, H10, H13, 52, CBR15 및 H14)의 절제된 뇌 조직으로부터 획득되었다. 환자 정보는 아래의 표 32에서 제공된다.
표 32: 환자 정보
먼저, 기관형적 해마 슬라이스가 단일 인간 TLE 환자로부터 획득되고, 시험관내에서 ACSF에서 11-12 일 동안 배양되고, 그리고 AAV9.GC(서열 번호: 101).GFP 벡터로 처리되었다. 상기 슬라이스는 차후에, 치상 과립 세포를 확인하기 위해 Prox1에 대해, 상기 벡터로 형질도입된 세포를 확인하기 위해 GFP에 대해 면역염색되었고, 그리고 Prox1 및 GFP 신호 사이의 중첩의 정도를 결정하기 위해 덧씌워졌다. 50% 초과의 치상 과립 세포가 GFP로 표지화되었고, 그리고 GFP 및 Prox1 사이에 실제적인 중첩이 관찰되었다 (도 23). 이들 결과는 RNA 올리고뉴클레오티드를 인코딩하는 AAV9 벡터가 인간 치상 과립 세포를 견실하게 형질감염시킬 수 있다는 것을 암시한다.
인간 기관형적 슬라이스가 Grik2 mRNA 표적화 안티센스 작제물 G9 (서열 번호: 68; 6명의 TLE 환자로부터 17개의 슬라이스) 또는 안티센스 작제물 GI (서열 번호: 77; 2명의 TLE 환자로부터 2개의 슬라이스)로 처리되었다. 벡터 처리된 슬라이스로부터 획득된 단백질 용해물에서 웨스턴 블롯이 수행되었다. GluK2 단백질 수준이 액틴에 대해 정규화되고 정량되었다. GluK2 단백질 발현의 녹다운이 G9로 처리된 인간 기관형적 해마 슬라이스의 5개 세트 중 5개에서 관찰되었다 (도 24). G9로 처리된 인간 슬라이스에서 수행된 예시적인 웨스턴 블롯이 도 25에서 도시되는데, 이것은 처리되지 않은 슬라이스와 비교하여 GluK2 수준에서 대략 40% 감소를 보여준다.
실시예 11: 인간 해마 슬라이스에서
Grik2
mRNA 표적화 이중-안티센스 발현 작제물의 시험관내 효능
인간 개체로부터 획득된 기관형적 해마 슬라이스를 이용하여, Grik2 mRNA 표적화, 이중-miRNA AAV 발현 벡터의 효능이 시험관내에서 검사되었다.
윤리학
인간 대뇌 조직을 이용한 실험을 위해, 모든 환자가 그들의 서면 동의를 제공하였고, 그리고 프로토콜이 CRB TBM/AP-HM (N° 271KAI)의 감독하에 INSERM (N° 2017-00031) 및 AP-HM (N° M17-06)에 의해 승인되었다.
뇌전증 환자로부터 획득된, 배양된 인간 해마 슬라이스:
인간 기관형적 슬라이스가 약제 내성 TLE로 진단된 4명의 환자 (11 - 58 세) (H pital de La Timone, Marseille, France)로부터 해마의 외과적 적출물로부터 준비되었다. 조직 블록이 차가운 (2-5℃), 산소화된 변형된 인공 뇌척수액 (mACSF)에 담겨 병원으로부터 실험실로 신중하게 수송되었고; mACSF는 132 콜린, 2.5 KCl, 1.25 NaH2PO4, 25 NaHCO3, 7 MgCl2, 0.5 CaCl2, 그리고 8 글루코오스 (mM로)를 내포하였고, 300-400 μm 두께 슬라이스가 동일한 용액에서 비브라톰 (Leica VT1200S)을 이용하여 생물안전 캐비닛 하에 준비되었다. 절단 후, 슬라이스가 동일자에 기록되거나 (급성 슬라이스), 또는 기록 전 시험관내에서 수일 동안 배양되었다 (기관형적 슬라이스, 하기 참조). 배양을 위해, 슬라이스가 실온 (22℃)에서 산소화된 "세척 매체"에서 15 분 동안 헹굼되었고; 세척 매체는 HEPES (20 mM)로 완성된 Hanks 균형 염 용액 (HBSS)을 내포하였다. 글루코오스 (17 mM) 및 항생제 (1% 항-항)가 전기생리학적 기록을 위해 첨가되었다. 기관형적 슬라이스가 6-웰 평판 (30 mm 트랜스웰) 내에 개별 세포 배양액 삽입물 (PICMORG50) 상에 배치되었다. 1 mL의 배양 배지가 각 웰에 떨어뜨려졌고; 배양 배지는 50% MEM, 25% 말 또는 인간 혈청, 15% HBSS, 2% B27, 0.5% 항생제, 11.8 mM 글루코오스, 그리고 20 mM 수크로오스를 내포하였다. 배양 평판이 37 ℃/5% CO2에서 인큐베이터 내에 유지되었다. 배양 배지가 2 일마다 교체되었고 첫 주 동안 항생제 (항-항)를 내포하였다. 기관형적 슬라이스의 전기생리학적 기록이 11 내지 15 DIV 후 수행되었다.
인간 기관형적 슬라이스의 바이러스 감염
감염을 위해, AAV9 작제물을 내포하는 PBS 배지가 DIV1에 인간 기관형적 슬라이스에 직접적으로 첨가되었다. 최종 바이러스 역가는 작제물 GC (서열 번호: 101), G9-S1 (서열 번호: 775) 및 DMTPV3 (서열 번호: 787), 각각에 대해 1.8E+10 GC/mL이었다. AAV9.hSyn.GFP 벡터가 G9-S1 및 DMTPV3 실험을 위한 대조로서 이용되었다. 검사된 벡터에 관한 상세한 정보는 아래의 표 33에서 제공된다.
표 33: miRNA 단일 및 이중 작제물에 대한 상세
전기생리학적 기록
인간 기관형적 슬라이스가 30-32℃에 유지된 기록 챔버로 개별적으로 이전되고, 그리고 산소화된 (95% O2 및 5% CO2) ACSF (생리학적 상태), 또는 5 μM 가바진 및 50 μM 4-AP를 내포하는 ACSF (과흥분 상태)로 연속적으로 관류되었다 (2-3 mL/분). 지역장 전위 (LFP) 기록이 치상회의 과립 세포 층에 배치된 단극 니크롬선으로 이루어졌다. LFP가 DAM-80 증폭기로 기록되었다 (저역 필터, 0.1 Hz; 고역 필터, 3 KHz; World Precision Instruments, Sarasota, FL); 데이터가 Digidata 1440A (Molecular Devices)를 이용하여 컴퓨터로 디지털화되고 (20 kHz), 그리고 Clampex 10.1 소프트웨어 (PClamp, Molecular Devices)를 이용하여 획득되었다. 신호가 Clampfit 9.2 (PClamp) 및 MiniAnalysis 6.0.1 (Synaptosoft, Decatur, GA)을 이용하여 오프라인 분석되었다. 뇌전증모양 방전 (ED)은 >30 ms의 지속 기간의 LFP를 갖는 멀티유닛 발사의 증가로 구성된다.
통계
모든 값은 평균 ± SEM으로서 제공된다. 통계학적 분석이 Graphpad Prism 7 (GraphPad Software, La Jolla, CA)을 이용하여 수행되었다. 집단간 비교를 위해, 미가공 데이터가 만 휘트니 검정에 의해 분석되었다. 유의성 수준은 p < 0.05로 설정되었다.
결과
Grik2 표적화 마이크로RNA 서열인 G9-S1 (서열 번호: 775)을 인코딩하는 AAV 발현 작제물, 스크램블 대조 서열인 GC (서열 번호: 101)를 인코딩하는 별개의 AAV 작제물, 그리고 Grik2를 표적으로 하는 이중 miRNA 작제물인 DMTPV3 (서열 번호: 787)을 인코딩하는 세 번째 벡터가 생리학적 상태에서 검사되었다 (방법 참조). 먼저, 과흥분 용액 (방법 참조)을 이용하여, 자연발생적 ED를 생성하는 네트워크의 기능성이 평가되었다; 진행 중인 자연발생적 ED와 반응하는 슬라이스가 선택되었다. 이들 슬라이스는 이후, 표준 생리학적 배지로 다시 전환되고 30 분 동안 세척되었다. 대조 조건 (예를 들면, GC-인코딩 작제물로 슬라이스의 감염)에서, 이들 슬라이스는 다수의 자연발생적 재발성 ED를 나타냈다 (0.3 ± 0.11 Hz, n=6개 슬라이스; 도 26a). 놀랍게도, 슬라이스가 G9-S1로 처리될 때, ED의 억제가 관찰되었다 (대략 93%; 0.019 ± 0.013 Hz, n=7개 슬라이스, p=0.0029, 만 휘트니 검정; 도 26b-26c, 표 34 참조).
표 34: 뇌전증모양 방전 - 평균
+
SEM
게다가, 이중 안티센스 벡터 DMTPV3으로 인간 기관형적 해마 슬라이스의 처리는 qRT-PCR에 의해 계측될 때, Grik2의 수준에서 실제적인 감소를 생성하였다 (도 27a). 생리학적 상태하에 (예를 들면, ACSF에서) 슬라이스에 투여될 때, DMTPV3은 AAV9.hSyn.GFP로 처리된 슬라이스 (도 27c-27d)와 비교하여, 뇌전증모양 방전에서 현저한 감소를 생성하였다 (도 27b).
이런 이유로, 이들 조사 결과는 Grik2 mRNA를 표적으로 하는 단일 및 이중 안티센스 서열을 인코딩하는 AAV 벡터에 의한 GluK2의 하향조절이 난치성 TLE를 겪는 환자로부터 획득된 해마 조직에서 뇌전증모양 활성을 억제하기 위한 효과적인 전략이라는 것을 증명한다.
실시예 12: 포장의 벡터 다중성의 분석
스터퍼 서열의 포함을 통해 벡터 길이를 증가시키는 영향이 평가되었다. 벡터 내로 통합된 스터퍼 (또는 충전) 서열은 유전자 전달 이후에 세포독성의 명백한 결여로 인해 선택되는 비코딩 서열이었다. 비코딩 서열 내에 전사 개시 또는 완료에 관련된 조절 요소의 제거는 스터퍼 서열이 전사 활성에 대하여 비활성이 되도록 만들었다.
다양한 유전체 크기의 벡터가 TapeStation 방법을 이용한 전기이동에 의해 평가되었다. DNA 분자의 분석이 D5000 시약 (Agilent #5067- 5593)을 이용한 높은 민감도 (HS) D5000 ScreenTape (Agilent #5067- 5592)에 대한 제조업체의 권장된 프로토콜에 따라서 수행되었다. TapeStation은 이중 가닥 (ds)DNA (단일 가닥 (ss)DNA보다는)를 계측하고, 그리고 TapeStation 평가는 염기쌍에서 동등한 근사치의 dsAAV 유전체 길이를 형성하는 2개의 상보성 ssAAV 유전체의 어닐링에 의존한다. 표 35에서 SMSPV1 (서열 번호: 818), SMSPV2 (서열 번호: 820) 및 SMSPV3 (서열 번호: 822)(SMSPV = 단일 마이크로RNA 단일 프로모터 벡터)로서 예시된, AAV의 포장 용량의 50% 미만을 갖는 유전체를 인코딩하는 벡터 (이들은 각각, 단일 miRNA 카세트를 인코딩한다)는 1.5-1.7 kb의 유전체 길이를 갖고, 그리고 캡시드마다 복수 (예를 들면, 2 또는 3개)의 유전체를 포장하는 잠재력을 갖는다. 따라서, SMSPV1, SMSPV2 및 SMSPV3 제조물로부터 "완전" 캡시드 중 단지 44%-61%만 단일 AAV 유전체를 내포한다. 이들 작제물 (각각, SMSPV4 (서열 번호: 804), SMSPV5 (서열 번호: 806) 및 SMSPV6 (서열 번호: 808)) 각각에 스터퍼 서열 (예를 들면, 서열 번호: 815 및/또는 서열 번호: 816)의 추가는 단일 AAV 유전체를 내포하는 각 벡터 프렙 내에 캡시드의 백분율을 72%-75%로 증가시킨다. 최종적으로, 2개의 구별되는 miRNA를 인코딩하는 연쇄체화된 유전체 (예를 들면, DMSPV1 (서열 번호: 812); DMSPV = 이중 마이크로RNA 단일 프로모터 벡터)에 스터퍼 서열의 추가는 87%의 단일 전장 AAV 유전체 내포 캡시드를 야기한다 (표 35 참조).
표 35: 스터퍼 서열이 있는 경우 및 없는 경우에 벡터의 유전체 분석
실시예 13: 생쥐 피질 뉴런에서 GluK2 발현의 시험관내 녹다운
출생후 0일 자 (P0)-P1 C57Bl6/J 생쥐로부터 해리된 피질 뉴런이 준비되었고 웰마다 5.5E+5개 세포의 농도로 6-웰 평판에 파종되었다. 도말 후 2 또는 3 일째 (생체내 일자, DIV2-3)에, 배지의 절반이 제거되었고, 그리고 바이러스 벡터가 MOI 7.5E+4로 추가되었다. DIV 13에, 생쥐 뉴런 배양액이 용해되었고, 그리고 용해물이 SDS-PAGE 및 면역블로팅에 이용되었다. 면역염색을 위해, 하기 항체가 적용되었다: 1:2000으로 희석된 토끼 항-GluK2/3 (클론 NL9 04-921; Merck-Millipore) 및 1:5000으로 희석된 생쥐 항- β-액틴 (A5316; Sigma)이 일차 항체로서 이용되었고, 그리고 1:15000으로 희석된, 염소에서 생산된 적합한 800 nm 형광단-접합된 이차 항체 (IRDye 800 염소 항생쥐 Li-COR 926-32210 또는 IRDye 800 염소 항토끼 Li-COR 926-32211)가 이차 항체로서 이용되었다. Li-COR 상에서 800 nm에서 형광을 계측함으로써, 표적 단백질이 검출되었다. 분석이 Empiria studio 소프트웨어로 수행되었다. 정량을 위해, 각 레인의 형광 신호의 강도가 β-액틴 발현에 의해, 이후 대조 조건에 의해 정규화되었다. 이들 실험 조건하에, 발현 작제물 DMTPV8 (서열 번호: 813) 및 DMSPV1 (서열 번호: 812)은 대조 벡터와 대비하여, 각각 GluK2 발현에서 73 ± 17% 및 71 ± 4%의 감소를 나타냈다 (평균 ± S.E.M.; 도 28 참조).
실시예 14: 측두엽 뇌전증의 생쥐 모형에서 GluK2의 생체내 녹다운
6-9 주령 Swiss 수컷 생쥐를 이용하여 실험이 수행되었다. 생쥐 (30-40 g)는 필로카르핀 (300-350 mg/kg)의 복막내 투여 30 분 전에 스코폴라민 (1 mg/kg)이 피하 (s.c.)로 제공되었다. 카페인 (40 mg/kg)이 첫 번째 발작 10 분 후에 투여되었다. 디아제팜 (10 mg/kg)이 뇌전증 지속증의 개시 1 시간 후에 투여되었다. AAV 투여를 위해, 뇌전증 생쥐 (뇌전증 지속증 후 >2 개월)는 이소플루란 마취하에 마취되었다 (100% O2 하에 유도의 경우 5% 및 유지의 경우 2%). 리도카인 (2%)의 국부 투여의 존재에서, 해마의 등면 및 복면 치상회 내로 AAV를 양측으로 주사하기 위해 4개의 홀이 천공되었다 (AP -1.8 mm, ML ±1 mm, DV - 2 mm, 그리고 AP -3.3 mm, ML ± 2.3 mm, DV - 2.5 mm). 각 주사를 위해, Hamilton 주입기가 AAV 전달에 이용되었다. 뇌 조직이 삽관 위를 미끄러져 가도록 하기 위한 5-분 지연 후, AAV를 내포하는 용액의 용적 = 1.0 μL/주사 부위 x 4 주사 부위가 천천히 주입되었다 (속도: 0.2 μL/분). 뇌전증 생쥐 (뇌전증 지속증 후 >2 개월)의 운동이 AAV 주사 1 주 전에 및 2 주 후에 평가되었다. 비-뇌전증 생쥐 (야생형 Swiss 수컷 생쥐, 18-21 주령)의 운동 역시 평가되었다. 생쥐가 환경에 익숙해지도록, 실험 하루 전에 행동 분석실로 이전되었다; 이들 생쥐는 실온 (20-22℃)에서 9:00-18:00 명암 주기로 유지되었고 사료와 물에 무제한 접근이 가능하였다. 검사된 동물과 접촉된 모든 재료는 그 후에, 후각 신호를 예방하기 위해 아세트산으로 세척되었다. 먼저, 자발적 탐색 행동이 전술된 오픈 필드 시험으로 검사되었다. 간단히 말하면, 생쥐가 10 분 동안 50 x 50 x 50 cm 청색 폴리염화비닐 상자의 중심에 배치되었고, 그리고 궤적이 추적 소프트웨어 EthoVision Color (Noldus, The Netherlands)에 연결된 비디오 카메라로 기록되었다; 10-분 자발적 탐색 동안 생쥐에 의해 커버된 속도 및 총 거리가 분석되었다.
DMTPV8 및 DMSPV1을 이용한 생쥐의 처리는 뇌전증유발성 행동에 대한 프록시인 과운동에서 감소를 야기하였다. 과운동에서 이러한 감소는 용량 의존성이었다. 양쪽 용량이 DMTPV8 및 DMSPV1에 대한 효능을 보여주긴 했지만, 3.6xE+9가 3.6xE+8보다 효과적이었다. 3.6xE+9의 높은 용량에서 DMSPV1이 검사된 모든 조건 중 가장 강한 효과를 나타냈다 (도 29).
실험의 별개 세트에서, 생쥐가 벡터 작제물 SMSPV4 (서열 번호: 804), SMSPV5 (서열 번호: 806), 또는 SMSPV6 (서열 번호: 808)으로 처리되었고, 그리고 운동이 전술된 바와 동일한 방식으로 검사되었다. 이들 작제물을 이용한 생쥐의 처리는 과운동 (뇌전증유발성 행동에 대한 프록시)에서 감소를 야기한 반면, 대조 벡터 (CV)를 이용한 처리는 어떤 영향도 주지 않았다 (도 31). mRNA 또는 단백질을 생산하지 않는 유전체를 내포하지만, 그 대신에 5' ITR에서 3' ITR로, RBG 폴리A (서열 번호: 751), CpG 고갈된 닭 베타 액틴 인트론 (서열 번호: 824), 그리고 서열 번호: 823의 비코딩 스터퍼 서열을 내포하는 AAV 캡시드를 모방하는 CV가 설계되었다. 상류 RBG 폴리A 서열은 다른 AAV 벡터와의 정확한 ddPCR 역가 비교를 가능하게 하고 5' ITR로부터 임의의 잠재적 전사를 제거한다. 항-Grik2 작제물을 이용한 처리 이후에 과운동에서 감소는 용량 의존성이었다. 양쪽 용량, 3.6xE+9 및 3.6xE+8이 SMSPV6에 대한 효능을 보여주긴 했지만, SMSPV4의 경우에 3.6xE+9 용량이 3.6xE+8보다 효과적이었다. SMSPV5의 경우에, 3.6xE+9 및 3.6xE+8 용량 둘 모두 유사한 효능을 보여주었다. SMSPV4는 검사된 모든 조건 중 3.6xE+9의 높은 용량에서 가장 강한 효과를 나타낸 반면, 3.6xE+8의 낮은 용량에서 SMSPV4는 과운동에 대한 어떤 영향도 주지 않았다 (도 31).
다른 구체예
설명된 개시의 다양한 변형과 변이는 발명의 범위와 사상으로부터 벗어나지 않으면서, 당업자에게 명백할 것이다. 비록 본원 발명이 특정한 구체예와 관련하여 설명되긴 했지만, 청구된 바와 같은 발명은 이런 특정한 구체예에 부당하게 한정되지 않는 것으로 이해되어야 한다. 실제로, 당업자에게 명백한, 본원 발명을 실행하기 위한 설명된 방식의 다양한 변형이 본원 발명의 범위 안에 있는 것으로 의도된다.
다른 구체예는 청구항에 있다.
참고문헌
본 출원의 전반에서, 다양한 참고문헌이 본원 발명과 관련되는 최신 기술을 설명한다. 이들 참고문헌의 개시는 본원에서 참조로서 본원 개시 내로 편입된다:
Bahn S., Volk B., Wisden W. (1994). Kainate receptor gene expression in the developing rat brain. J. Neurosci. 14 5525-5547. 10.1523/JNEUROSCI.14-09-05525.1994.
Boudreau Ryan L., Rodrguez-Lebrn Edgardo, Davidson Beverly L., RNAi medicine for the brain: progresses and challenges, Human Molecular Genetics, Volume 20, Issue R1, 15 April 2011, Pages R21-R27
Bouvier G., Larsen R. S., Rodrguez-Moreno A., Paulsen O., Sjostrom P. J. (2018). Towards resolving the presynaptic NMDA receptor debate. Curr. Opin. Neurobiol. 51 1-7. 10.1016/j.conb.2017.12.020
Crpel V, Mulle C (2015) Physiopathology of kainate receptors in epilepsy. Curr Opin Pharmacol 20:83-88; doi: 10.1016/j.coph.2014.11.012. Epub 2014 Dec 13.
Englot, DJ., et al (2013) Seizure outcomes after resective surgery for extra-temporal lobe epilepsy in pediatric patients: A systematic review. J. Neurosurgery. 12(2):97-201
Fritsch B., Reis J., Gasior M., Kaminski R. M., Michael A., Rogawski M. A. (2014). Role of GluK1 kainate receptors in seizures, epileptic discharges, and epileptogenesis. J. Neurosci. 34 5765-5775. 10.1523/JNEUROSCI.5307-13.2014
Gabriel S, Njunting M, Pomper JK, Merschhemke M, Sanabria ERG, Eilers A, Kivi A, Zeller M, Meencke H-J, Cavalheiro E a, Heinemann U, Lehmann T-N (2004) Stimulus and potassium-induced epileptiform activity in the human dentate gyrus from patients with and without hippocampal sclerosis. J Neurosci 24:10416-10430.
Gruber A., Lorenz R., Bernhart S.H., Neubck R., Hofacker I.L (2008). The Vienna RNA Websuite. Nucleic Acids Research. 36:W70-4
Hardy J. (2010). Genetic analysis of pathways to Parkinson disease. Neuron, 68(2), 201-206. doi:10.1016/j.neuron.2010.10.014
Melyan Z., Lancaster B., Wheal H. V. (2004). Metabotropic regulation of intrinsic excitability by synaptic activation of kainate receptors. J. Neurosci. 24 4530-4534. 10.1523/JNEUROSCI.5356-03.2004
Melyan Z., Wheal H. V., Lancaster B. (2002). Metabotropic-mediated kainate receptor regulation of isAHP and excitability in pyramidal cells. Neuron 34 107-114. 10.1016/S0896-6273(02)00624-4
Mulle C., Sailer A., Prez-Otao I., Dickinson-Anson H., Castillo P. E., Bureau I., et al. (1998). Altered synaptic physiology and reduced susceptibility to kainate-induced seizures in GluR6-deficient mice. Nature 392 601-605.
Peret A, Christie L a., Ouedraogo DW, Gorlewicz A, Epsztein J, Mulle C, Crpel V (2014) Contribution of Aberrant GluK2-Containing Kainate Receptors to Chronic Seizures in Temporal Lobe Epilepsy. Cell Rep 8:347-354.
Reiner A, Arant RJ, and Isacoff EY (2012) Assembly Stoichiometry of the GluK2/GluK5 Kainate Receptor Complex. Cell Rep 1:234-240.
Represa A, Le Gall La Salle G, Ben-Ari Y (1989a) Hippocampal plasticity in the kindling model of epilepsy in rats. Neurosci Lett 99:345-350.
Represa A, Robain O, Tremblay E, Ben-Ari Y (1989b) Hippocampal plasticity in childhood epilepsy. Neurosci Lett 99:351-355.
Rodrguez-Moreno A., Herreras O., Lerma J. (1997). Kainate receptors presynaptically downregulate GABAergic inhibition in the rat hippocampus. Neuron 19 893-901. 10.1016/S0896-6273(00)80970-8.
Rodrguez-Moreno A., Sihra T. S. (2007a). Kainate receptors with a metabotropic modus operandi. Trends Neurosci. 30 630-637.
Rodrguez-Moreno A., Sihra T. S. (2007b). Metabotropic actions of kainate receptors in the CNS. J. Neurochem. 103 2121-2135.
Sapru Mohan K., Yates Jonathan W., Hogan Shea, Jiang Lixin, Halter Jeremy, Bohn Martha C. (2006). Silencing of human α-synuclein in vitro and in rat brain using lentiviral-mediated RNAi. Neurology.198:382-390
Smolders I., Bortolotto Z. A., Clarke V. R., Warre R., Khan G. M., O'Neill M. J., et al. (2002). Antagonists of GLU(K5)-containing kainate receptors prevent pilocarpine-induced limbic seizures. Nat. Neurosci. 5 796-804. 10.1038/nn88
Sutula T, Cascino G, Cavazos J, Parada I, Ramirez L (1989) Mossy fiber synaptic reorganization in the epileptic human temporal lobe. Ann Neurol 26:321-330.
Tauck DL, Nadler J V (1985) Evidence of functional mossy fiber sprouting in hippocampal formation of kainic acid-treated rats. J Neurosci 5:1016-1022
Valbuena S., Lerma J. (2016). Non-canonical signaling, the hidden life of ligand-gated ion channels. Neuron 92 316-329. 10.1016/j.neuron.2016.10.016
Wang L., Bai J., and Hu Y. (2007) Identification of the RA Response Element and Transcriptional Silencer in Human alphaCaMKII Promoter. Mol. Biol. Rep. 35(1):37-44
Zinn, E., Pacouret, S., Khaychuk, V., Turunen, H. T., Carvalho, L. S., Andres-Mateos, E., … Vandenberghe, L. H. (2015). In Silico Reconstruction of the Viral Evolutionary Lineage Yields a Potent Gene Therapy Vector. Cell reports, 12(6), 1056-1068. doi:10.1016/j.celrep.2015.07.019
SEQUENCE LISTING
<110> INSERM (INSTITUT NATIONAL DE LA SANTE ET DE LA RECHERCHE
MEDICALE)
UNIVERSITE D'AIX MARSEILLE
UNIVERSITE DE BORDEAUX
REGENXBIO INC.
CENTRE NATIONAL DE LA RECHERCHE SCIENTIFIQUE
CORLIEVE THERAPEUTICS
<120> METHODS AND COMPOSITIONS FOR TREATING EPILEPSY
<130> 51460-003WO4
<150> US 63/185,699
<151> 2021-05-07
<150> US 63/137,669
<151> 2021-01-14
<150> US 63/050,742
<151> 2020-07-10
<160> 824
<170> PatentIn version 3.5
<210> 1
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 1
aaggguagua uuggguagca a 21
<210> 2
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 2
aggcccgaag auggcagcca c 21
<210> 3
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 3
agcauugcag auggacugca c 21
<210> 4
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 4
uguuaacacc acuguaucgg u 21
<210> 5
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 5
aaucggguuu cggaggugcc u 21
<210> 6
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 6
aucggguuuc ggaggugccu g 21
<210> 7
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 7
uguagauaac ucucugagga g 21
<210> 8
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 8
uuaguucacg aaccauucca u 21
<210> 9
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 9
aauaacuggg gccuguggcu u 21
<210> 10
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 10
uuagugacac uuuuauuggu u 21
<210> 11
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 11
uuauuauuug ggauaugggg g 21
<210> 12
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 12
gauguucgcu ggcuuucccu u 21
<210> 13
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 13
uguuccacug ucagaaaggc g 21
<210> 14
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 14
cguuccacug ucagaaaggc g 21
<210> 15
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 15
ugauauucca ucuccuggca a 21
<210> 16
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 16
auaagugaug aacaucugcu u 21
<210> 17
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 17
uauuuccuug auaauuggca u 21
<210> 18
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 18
uauccucaau ucuucuacca u 21
<210> 19
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 19
cauccucaau ucuucuacca u 21
<210> 20
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 20
ugucgauuac acugcaagga a 21
<210> 21
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 21
gaugcgucga guggugaccg c 21
<210> 22
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 22
cucgaacaua gguaauagcc a 21
<210> 23
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 23
gguucgggua gaaaugagga u 21
<210> 24
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 24
uuagcguucg gcuccugggu u 21
<210> 25
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 25
cuagcguucg gcuccugggu u 21
<210> 26
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 26
guucggcucc uggguucggg u 21
<210> 27
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 27
aacguuggug gugcacacgc a 21
<210> 28
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 28
uggugcgccu gaagacugga u 21
<210> 29
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 29
cggugcgccu gaagacugga u 21
<210> 30
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 30
uagcgggucu guaugugggg a 21
<210> 31
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 31
cagcgggucu guaugugggg a 21
<210> 32
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 32
uacgcacuac cauucaugcu u 21
<210> 33
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 33
cacgcacuac cauucaugcu u 21
<210> 34
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 34
uucgaugguu guugacucca u 21
<210> 35
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 35
cucgaugguu guugacucca u 21
<210> 36
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 36
agcgggucug uaugugggga a 21
<210> 37
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 37
uugaacggcc acagacacca c 21
<210> 38
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 38
aaagcggguc ccgaagcgcc a 21
<210> 39
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 39
agacgccugg guuuguacca u 21
<210> 40
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 40
aaacgaauga gaccagugcu g 21
<210> 41
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 41
auaaacgcag uccacuucca a 21
<210> 42
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 42
gaccgcagac acgaucacgg c 21
<210> 43
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 43
uucggcuccu ggguucgggu a 21
<210> 44
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 44
uaaagcgggu cccgaagcgc c 21
<210> 45
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 45
uacggcaccc acuuccccga u 21
<210> 46
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 46
cacggcaccc acuuccccga u 21
<210> 47
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 47
agcgccaggg uuuaugucga u 21
<210> 48
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 48
gucuccgcuu cccaaaccca u 21
<210> 49
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 49
gacgacaguu ugugcuuggg u 21
<210> 50
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 50
agccucgugg aaaccagggg u 21
<210> 51
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 51
ugucucgaua uggagaaccc a 21
<210> 52
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 52
ugacgcuggc acuucaggga c 21
<210> 53
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 53
cgacgcuggc acuucaggga c 21
<210> 54
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 54
agacacgauc acggcauggu c 21
<210> 55
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 55
uuccccgauc uagcguucgg c 21
<210> 56
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 56
ugcaaucguu ccaucgacca c 21
<210> 57
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 57
uugaaucggg uuucggaggu g 21
<210> 58
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 58
cugaaucggg uuucggaggu g 21
<210> 59
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 59
aaggcccgaa gauggcagcc a 21
<210> 60
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 60
uugagucgaa gauuauaccu u 21
<210> 61
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 61
ggcuaguaac aucaucaccu c 21
<210> 62
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 62
agauaucagg ggagagagga u 21
<210> 63
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 63
gguugcauau uuccacagga a 21
<210> 64
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 64
ugcgucgagu ggugaccgca g 21
<210> 65
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 65
uuagucggag agcauccggg a 21
<210> 66
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 66
augcgucgag uggugaccgc a 21
<210> 67
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 67
uauccgggag aaauccagca c 21
<210> 68
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 68
cccauagcua augccuguuu u 21
<210> 69
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 69
uugucaucau ucccauagcu a 21
<210> 70
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 70
cauucccaua gcuaaugccu g 21
<210> 71
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 71
cccauagcua augccugcuu u 21
<210> 72
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 72
aaaccaccaa augccuccca c 21
<210> 73
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 73
augauaagug ugaaaaacca c 21
<210> 74
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 74
agucgaugac cuucucucga a 21
<210> 75
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 75
uucgaacaua gguaauagcc a 21
<210> 76
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 76
gacaccuggu gcuuccagcg g 21
<210> 77
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 77
gucucgauau ggagaaccca u 21
<210> 78
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 78
ugauauggag aacccauggg a 21
<210> 79
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 79
gauauggaga acccauggga g 21
<210> 80
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 80
cagagcauug cagauggacu g 21
<210> 81
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 81
cccagagcau ugcagaugga c 21
<210> 82
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 82
uaccacgucu gagucagggu u 21
<210> 83
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 83
caccacgucu gagucagggu u 21
<210> 84
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 84
uugagucagg guugcaaggg u 21
<210> 85
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 85
agagcuccaa cuccaaacca g 21
<210> 86
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 86
ugauggagcu uugaugagcu c 21
<210> 87
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 87
uauagguaau agccagugga g 21
<210> 88
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 88
cauagguaau agccagugga g 21
<210> 89
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 89
gagcaacugc aaggucagcu u 21
<210> 90
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 90
agguaauagc caguggagca a 21
<210> 91
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 91
agucgucaua aauccaucca g 21
<210> 92
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 92
acugacauag aaggaaucuu u 21
<210> 93
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 93
uagagacuga cauagaagga a 21
<210> 94
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 94
ugucauaaau ccauccagca a 21
<210> 95
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 95
uaucagucgu cauaaaucca u 21
<210> 96
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 96
uucauaugua aagccaagga u 21
<210> 97
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 97
acugacauag aaggaauccu u 21
<210> 98
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 98
aauggacaau ggaauggaau g 21
<210> 99
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 99
auggaaugga augguucgug a 21
<210> 100
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 100
uagucggaga gcauccggga g 21
<210> 101
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 101
uaauguuagu cauguccacc g 21
<210> 102
<211> 908
<212> PRT
<213> Homo sapiens
<400> 102
Met Lys Ile Ile Phe Pro Ile Leu Ser Asn Pro Val Phe Arg Arg Thr
1 5 10 15
Val Lys Leu Leu Leu Cys Leu Leu Trp Ile Gly Tyr Ser Gln Gly Thr
20 25 30
Thr His Val Leu Arg Phe Gly Gly Ile Phe Glu Tyr Val Glu Ser Gly
35 40 45
Pro Met Gly Ala Glu Glu Leu Ala Phe Arg Phe Ala Val Asn Thr Ile
50 55 60
Asn Arg Asn Arg Thr Leu Leu Pro Asn Thr Thr Leu Thr Tyr Asp Thr
65 70 75 80
Gln Lys Ile Asn Leu Tyr Asp Ser Phe Glu Ala Ser Lys Lys Ala Cys
85 90 95
Asp Gln Leu Ser Leu Gly Val Ala Ala Ile Phe Gly Pro Ser His Ser
100 105 110
Ser Ser Ala Asn Ala Val Gln Ser Ile Cys Asn Ala Leu Gly Val Pro
115 120 125
His Ile Gln Thr Arg Trp Lys His Gln Val Ser Asp Asn Lys Asp Ser
130 135 140
Phe Tyr Val Ser Leu Tyr Pro Asp Phe Ser Ser Leu Ser Arg Ala Ile
145 150 155 160
Leu Asp Leu Val Gln Phe Phe Lys Trp Lys Thr Val Thr Val Val Tyr
165 170 175
Asp Asp Ser Thr Gly Leu Ile Arg Leu Gln Glu Leu Ile Lys Ala Pro
180 185 190
Ser Arg Tyr Asn Leu Arg Leu Lys Ile Arg Gln Leu Pro Ala Asp Thr
195 200 205
Lys Asp Ala Lys Pro Leu Leu Lys Glu Met Lys Arg Gly Lys Glu Phe
210 215 220
His Val Ile Phe Asp Cys Ser His Glu Met Ala Ala Gly Ile Leu Lys
225 230 235 240
Gln Ala Leu Ala Met Gly Met Met Thr Glu Tyr Tyr His Tyr Ile Phe
245 250 255
Thr Thr Leu Asp Leu Phe Ala Leu Asp Val Glu Pro Tyr Arg Tyr Ser
260 265 270
Gly Val Asn Met Thr Gly Phe Arg Ile Leu Asn Thr Glu Asn Thr Gln
275 280 285
Val Ser Ser Ile Ile Glu Lys Trp Ser Met Glu Arg Leu Gln Ala Pro
290 295 300
Pro Lys Pro Asp Ser Gly Leu Leu Asp Gly Phe Met Thr Thr Asp Ala
305 310 315 320
Ala Leu Met Tyr Asp Ala Val His Val Val Ser Val Ala Val Gln Gln
325 330 335
Phe Pro Gln Met Thr Val Ser Ser Leu Gln Cys Asn Arg His Lys Pro
340 345 350
Trp Arg Phe Gly Thr Arg Phe Met Ser Leu Ile Lys Glu Ala His Trp
355 360 365
Glu Gly Leu Thr Gly Arg Ile Thr Phe Asn Lys Thr Asn Gly Leu Arg
370 375 380
Thr Asp Phe Asp Leu Asp Val Ile Ser Leu Lys Glu Glu Gly Leu Glu
385 390 395 400
Lys Ile Gly Thr Trp Asp Pro Ala Ser Gly Leu Asn Met Thr Glu Ser
405 410 415
Gln Lys Gly Lys Pro Ala Asn Ile Thr Asp Ser Leu Ser Asn Arg Ser
420 425 430
Leu Ile Val Thr Thr Ile Leu Glu Glu Pro Tyr Val Leu Phe Lys Lys
435 440 445
Ser Asp Lys Pro Leu Tyr Gly Asn Asp Arg Phe Glu Gly Tyr Cys Ile
450 455 460
Asp Leu Leu Arg Glu Leu Ser Thr Ile Leu Gly Phe Thr Tyr Glu Ile
465 470 475 480
Arg Leu Val Glu Asp Gly Lys Tyr Gly Ala Gln Asp Asp Ala Asn Gly
485 490 495
Gln Trp Asn Gly Met Val Arg Glu Leu Ile Asp His Lys Ala Asp Leu
500 505 510
Ala Val Ala Pro Leu Ala Ile Thr Tyr Val Arg Glu Lys Val Ile Asp
515 520 525
Phe Ser Lys Pro Phe Met Thr Leu Gly Ile Ser Ile Leu Tyr Arg Lys
530 535 540
Pro Asn Gly Thr Asn Pro Gly Val Phe Ser Phe Leu Asn Pro Leu Ser
545 550 555 560
Pro Asp Ile Trp Met Tyr Ile Leu Leu Ala Tyr Leu Gly Val Ser Cys
565 570 575
Val Leu Phe Val Ile Ala Arg Phe Ser Pro Tyr Glu Trp Tyr Asn Pro
580 585 590
His Pro Cys Asn Pro Asp Ser Asp Val Val Glu Asn Asn Phe Thr Leu
595 600 605
Leu Asn Ser Phe Trp Phe Gly Val Gly Ala Leu Met Gln Gln Gly Ser
610 615 620
Glu Leu Met Pro Lys Ala Leu Ser Thr Arg Ile Val Gly Gly Ile Trp
625 630 635 640
Trp Phe Phe Thr Leu Ile Ile Ile Ser Ser Tyr Thr Ala Asn Leu Ala
645 650 655
Ala Phe Leu Thr Val Glu Arg Met Glu Ser Pro Ile Asp Ser Ala Asp
660 665 670
Asp Leu Ala Lys Gln Thr Lys Ile Glu Tyr Gly Ala Val Glu Asp Gly
675 680 685
Ala Thr Met Thr Phe Phe Lys Lys Ser Lys Ile Ser Thr Tyr Asp Lys
690 695 700
Met Trp Ala Phe Met Ser Ser Arg Arg Gln Ser Val Leu Val Lys Ser
705 710 715 720
Asn Glu Glu Gly Ile Gln Arg Val Leu Thr Ser Asp Tyr Ala Phe Leu
725 730 735
Met Glu Ser Thr Thr Ile Glu Phe Val Thr Gln Arg Asn Cys Asn Leu
740 745 750
Thr Gln Ile Gly Gly Leu Ile Asp Ser Lys Gly Tyr Gly Val Gly Thr
755 760 765
Pro Met Gly Ser Pro Tyr Arg Asp Lys Ile Thr Ile Ala Ile Leu Gln
770 775 780
Leu Gln Glu Glu Gly Lys Leu His Met Met Lys Glu Lys Trp Trp Arg
785 790 795 800
Gly Asn Gly Cys Pro Glu Glu Glu Ser Lys Glu Ala Ser Ala Leu Gly
805 810 815
Val Gln Asn Ile Gly Gly Ile Phe Ile Val Leu Ala Ala Gly Leu Val
820 825 830
Leu Ser Val Phe Val Ala Val Gly Glu Phe Leu Tyr Lys Ser Lys Lys
835 840 845
Asn Ala Gln Leu Glu Lys Arg Ser Phe Cys Ser Ala Met Val Glu Glu
850 855 860
Leu Arg Met Ser Leu Lys Cys Gln Arg Arg Leu Lys His Lys Pro Gln
865 870 875 880
Ala Pro Val Ile Val Lys Thr Glu Glu Val Ile Asn Met His Thr Phe
885 890 895
Asn Asp Arg Arg Leu Pro Gly Lys Glu Thr Met Ala
900 905
<210> 103
<211> 869
<212> PRT
<213> Homo sapiens
<400> 103
Met Lys Ile Ile Phe Pro Ile Leu Ser Asn Pro Val Phe Arg Arg Thr
1 5 10 15
Val Lys Leu Leu Leu Cys Leu Leu Trp Ile Gly Tyr Ser Gln Gly Thr
20 25 30
Thr His Val Leu Arg Phe Gly Gly Ile Phe Glu Tyr Val Glu Ser Gly
35 40 45
Pro Met Gly Ala Glu Glu Leu Ala Phe Arg Phe Ala Val Asn Thr Ile
50 55 60
Asn Arg Asn Arg Thr Leu Leu Pro Asn Thr Thr Leu Thr Tyr Asp Thr
65 70 75 80
Gln Lys Ile Asn Leu Tyr Asp Ser Phe Glu Ala Ser Lys Lys Ala Cys
85 90 95
Asp Gln Leu Ser Leu Gly Val Ala Ala Ile Phe Gly Pro Ser His Ser
100 105 110
Ser Ser Ala Asn Ala Val Gln Ser Ile Cys Asn Ala Leu Gly Val Pro
115 120 125
His Ile Gln Thr Arg Trp Lys His Gln Val Ser Asp Asn Lys Asp Ser
130 135 140
Phe Tyr Val Ser Leu Tyr Pro Asp Phe Ser Ser Leu Ser Arg Ala Ile
145 150 155 160
Leu Asp Leu Val Gln Phe Phe Lys Trp Lys Thr Val Thr Val Val Tyr
165 170 175
Asp Asp Ser Thr Gly Leu Ile Arg Leu Gln Glu Leu Ile Lys Ala Pro
180 185 190
Ser Arg Tyr Asn Leu Arg Leu Lys Ile Arg Gln Leu Pro Ala Asp Thr
195 200 205
Lys Asp Ala Lys Pro Leu Leu Lys Glu Met Lys Arg Gly Lys Glu Phe
210 215 220
His Val Ile Phe Asp Cys Ser His Glu Met Ala Ala Gly Ile Leu Lys
225 230 235 240
Gln Ala Leu Ala Met Gly Met Met Thr Glu Tyr Tyr His Tyr Ile Phe
245 250 255
Thr Thr Leu Asp Leu Phe Ala Leu Asp Val Glu Pro Tyr Arg Tyr Ser
260 265 270
Gly Val Asn Met Thr Gly Phe Arg Ile Leu Asn Thr Glu Asn Thr Gln
275 280 285
Val Ser Ser Ile Ile Glu Lys Trp Ser Met Glu Arg Leu Gln Ala Pro
290 295 300
Pro Lys Pro Asp Ser Gly Leu Leu Asp Gly Phe Met Thr Thr Asp Ala
305 310 315 320
Ala Leu Met Tyr Asp Ala Val His Val Val Ser Val Ala Val Gln Gln
325 330 335
Phe Pro Gln Met Thr Val Ser Ser Leu Gln Cys Asn Arg His Lys Pro
340 345 350
Trp Arg Phe Gly Thr Arg Phe Met Ser Leu Ile Lys Glu Ala His Trp
355 360 365
Glu Gly Leu Thr Gly Arg Ile Thr Phe Asn Lys Thr Asn Gly Leu Arg
370 375 380
Thr Asp Phe Asp Leu Asp Val Ile Ser Leu Lys Glu Glu Gly Leu Glu
385 390 395 400
Lys Ile Gly Thr Trp Asp Pro Ala Ser Gly Leu Asn Met Thr Glu Ser
405 410 415
Gln Lys Gly Lys Pro Ala Asn Ile Thr Asp Ser Leu Ser Asn Arg Ser
420 425 430
Leu Ile Val Thr Thr Ile Leu Glu Glu Pro Tyr Val Leu Phe Lys Lys
435 440 445
Ser Asp Lys Pro Leu Tyr Gly Asn Asp Arg Phe Glu Gly Tyr Cys Ile
450 455 460
Asp Leu Leu Arg Glu Leu Ser Thr Ile Leu Gly Phe Thr Tyr Glu Ile
465 470 475 480
Arg Leu Val Glu Asp Gly Lys Tyr Gly Ala Gln Asp Asp Ala Asn Gly
485 490 495
Gln Trp Asn Gly Met Val Arg Glu Leu Ile Asp His Lys Ala Asp Leu
500 505 510
Ala Val Ala Pro Leu Ala Ile Thr Tyr Val Arg Glu Lys Val Ile Asp
515 520 525
Phe Ser Lys Pro Phe Met Thr Leu Gly Ile Ser Ile Leu Tyr Arg Lys
530 535 540
Pro Asn Gly Thr Asn Pro Gly Val Phe Ser Phe Leu Asn Pro Leu Ser
545 550 555 560
Pro Asp Ile Trp Met Tyr Ile Leu Leu Ala Tyr Leu Gly Val Ser Cys
565 570 575
Val Leu Phe Val Ile Ala Arg Phe Ser Pro Tyr Glu Trp Tyr Asn Pro
580 585 590
His Pro Cys Asn Pro Asp Ser Asp Val Val Glu Asn Asn Phe Thr Leu
595 600 605
Leu Asn Ser Phe Trp Phe Gly Val Gly Ala Leu Met Gln Gln Gly Ser
610 615 620
Glu Leu Met Pro Lys Ala Leu Ser Thr Arg Ile Val Gly Gly Ile Trp
625 630 635 640
Trp Phe Phe Thr Leu Ile Ile Ile Ser Ser Tyr Thr Ala Asn Leu Ala
645 650 655
Ala Phe Leu Thr Val Glu Arg Met Glu Ser Pro Ile Asp Ser Ala Asp
660 665 670
Asp Leu Ala Lys Gln Thr Lys Ile Glu Tyr Gly Ala Val Glu Asp Gly
675 680 685
Ala Thr Met Thr Phe Phe Lys Lys Ser Lys Ile Ser Thr Tyr Asp Lys
690 695 700
Met Trp Ala Phe Met Ser Ser Arg Arg Gln Ser Val Leu Val Lys Ser
705 710 715 720
Asn Glu Glu Gly Ile Gln Arg Val Leu Thr Ser Asp Tyr Ala Phe Leu
725 730 735
Met Glu Ser Thr Thr Ile Glu Phe Val Thr Gln Arg Asn Cys Asn Leu
740 745 750
Thr Gln Ile Gly Gly Leu Ile Asp Ser Lys Gly Tyr Gly Val Gly Thr
755 760 765
Pro Met Gly Ser Pro Tyr Arg Asp Lys Ile Thr Ile Ala Ile Leu Gln
770 775 780
Leu Gln Glu Glu Gly Lys Leu His Met Met Lys Glu Lys Trp Trp Arg
785 790 795 800
Gly Asn Gly Cys Pro Glu Glu Glu Ser Lys Glu Ala Ser Ala Leu Gly
805 810 815
Val Gln Asn Ile Gly Gly Ile Phe Ile Val Leu Ala Ala Gly Leu Val
820 825 830
Leu Ser Val Phe Val Ala Val Gly Glu Phe Leu Tyr Lys Ser Lys Lys
835 840 845
Asn Ala Gln Leu Glu Lys Glu Ser Ser Ile Trp Leu Val Pro Pro Tyr
850 855 860
His Pro Asp Thr Val
865
<210> 104
<211> 584
<212> PRT
<213> Homo sapiens
<400> 104
Met Lys Ile Ile Phe Pro Ile Leu Ser Asn Pro Val Phe Arg Arg Thr
1 5 10 15
Val Lys Leu Leu Leu Cys Leu Leu Trp Ile Gly Tyr Ser Gln Gly Thr
20 25 30
Thr His Val Leu Arg Phe Gly Gly Ile Phe Glu Tyr Val Glu Ser Gly
35 40 45
Pro Met Gly Ala Glu Glu Leu Ala Phe Arg Phe Ala Val Asn Thr Ile
50 55 60
Asn Arg Asn Arg Thr Leu Leu Pro Asn Thr Thr Leu Thr Tyr Asp Thr
65 70 75 80
Gln Lys Ile Asn Leu Tyr Asp Ser Phe Glu Ala Ser Lys Lys Ala Cys
85 90 95
Asp Gln Leu Ser Leu Gly Val Ala Ala Ile Phe Gly Pro Ser His Ser
100 105 110
Ser Ser Ala Asn Ala Val Gln Ser Ile Cys Asn Ala Leu Gly Val Pro
115 120 125
His Ile Gln Thr Arg Trp Lys His Gln Val Ser Asp Asn Lys Asp Ser
130 135 140
Phe Tyr Val Ser Leu Tyr Pro Asp Phe Ser Ser Leu Ser Arg Ala Ile
145 150 155 160
Leu Asp Leu Val Gln Phe Phe Lys Trp Lys Thr Val Thr Val Val Tyr
165 170 175
Asp Asp Ser Thr Gly Leu Ile Arg Leu Gln Glu Leu Ile Lys Ala Pro
180 185 190
Ser Arg Tyr Asn Leu Arg Leu Lys Ile Arg Gln Leu Pro Ala Asp Thr
195 200 205
Lys Asp Ala Lys Pro Leu Leu Lys Glu Met Lys Arg Gly Lys Glu Phe
210 215 220
His Val Ile Phe Asp Cys Ser His Glu Met Ala Ala Gly Ile Leu Lys
225 230 235 240
Gln Ala Leu Ala Met Gly Met Met Thr Glu Tyr Tyr His Tyr Ile Phe
245 250 255
Thr Thr Leu Asp Leu Phe Ala Leu Asp Val Glu Pro Tyr Arg Tyr Ser
260 265 270
Gly Val Asn Met Thr Gly Phe Arg Ile Leu Asn Thr Glu Asn Thr Gln
275 280 285
Val Ser Ser Ile Ile Glu Lys Trp Ser Met Glu Arg Leu Gln Ala Pro
290 295 300
Pro Lys Pro Asp Ser Gly Leu Leu Asp Gly Phe Met Thr Thr Asp Ala
305 310 315 320
Ala Leu Met Tyr Asp Ala Val His Val Val Ser Val Ala Val Gln Gln
325 330 335
Phe Pro Gln Met Thr Val Ser Ser Leu Gln Cys Asn Arg His Lys Pro
340 345 350
Trp Arg Phe Gly Thr Arg Phe Met Ser Leu Ile Lys Glu Ala His Trp
355 360 365
Glu Gly Leu Thr Gly Arg Ile Thr Phe Asn Lys Thr Asn Gly Leu Arg
370 375 380
Thr Asp Phe Asp Leu Asp Val Ile Ser Leu Lys Glu Glu Gly Leu Glu
385 390 395 400
Lys Ile Gly Thr Trp Asp Pro Ala Ser Gly Leu Asn Met Thr Glu Ser
405 410 415
Gln Lys Gly Lys Pro Ala Asn Ile Thr Asp Ser Leu Ser Asn Arg Ser
420 425 430
Leu Ile Val Thr Thr Ile Leu Glu Glu Pro Tyr Val Leu Phe Lys Lys
435 440 445
Ser Asp Lys Pro Leu Tyr Gly Asn Asp Arg Phe Glu Gly Tyr Cys Ile
450 455 460
Asp Leu Leu Arg Glu Leu Ser Thr Ile Leu Gly Phe Thr Tyr Glu Ile
465 470 475 480
Arg Leu Val Glu Asp Gly Lys Tyr Gly Ala Gln Asp Asp Ala Asn Gly
485 490 495
Gln Trp Asn Gly Met Val Arg Glu Leu Ile Asp His Lys Ala Asp Leu
500 505 510
Ala Val Ala Pro Leu Ala Ile Thr Tyr Val Arg Glu Lys Val Ile Asp
515 520 525
Phe Ser Lys Pro Phe Met Thr Leu Gly Ile Ser Ile Leu Tyr Arg Lys
530 535 540
Pro Asn Gly Thr Asn Pro Gly Val Phe Ser Phe Leu Asn Pro Leu Ser
545 550 555 560
Pro Asp Ile Trp Met Tyr Ile Leu Leu Ala Tyr Leu Gly Val Ser Cys
565 570 575
Val Leu Phe Val Ile Ala Arg Phe
580
<210> 105
<211> 832
<212> PRT
<213> Homo sapiens
<400> 105
Met Lys Ile Ile Phe Pro Ile Leu Ser Asn Pro Val Phe Arg Arg Thr
1 5 10 15
Val Lys Leu Leu Leu Cys Leu Leu Trp Ile Gly Tyr Ser Gln Gly Thr
20 25 30
Thr His Val Leu Arg Phe Gly Gly Ile Phe Glu Tyr Val Glu Ser Gly
35 40 45
Pro Met Gly Ala Glu Glu Leu Ala Phe Arg Phe Ala Val Asn Thr Ile
50 55 60
Asn Arg Asn Arg Thr Leu Leu Pro Asn Thr Thr Leu Thr Tyr Asp Thr
65 70 75 80
Gln Lys Ile Asn Leu Tyr Asp Ser Phe Glu Ala Ser Lys Lys Ala Cys
85 90 95
Asp Gln Leu Ser Leu Gly Val Ala Ala Ile Phe Gly Pro Ser His Ser
100 105 110
Ser Ser Ala Asn Ala Val Gln Ser Ile Cys Asn Ala Leu Gly Val Pro
115 120 125
His Ile Gln Thr Arg Trp Lys His Gln Val Ser Asp Asn Lys Asp Ser
130 135 140
Phe Tyr Val Ser Leu Tyr Pro Asp Phe Ser Ser Leu Ser Arg Ala Ile
145 150 155 160
Leu Asp Leu Val Gln Phe Phe Lys Trp Lys Thr Val Thr Val Val Tyr
165 170 175
Asp Asp Ser Thr Gly Leu Ile Arg Leu Gln Glu Leu Ile Lys Ala Pro
180 185 190
Ser Arg Tyr Asn Leu Arg Leu Lys Ile Arg Gln Leu Pro Ala Asp Thr
195 200 205
Lys Asp Ala Lys Pro Leu Leu Lys Glu Met Lys Arg Gly Lys Glu Phe
210 215 220
His Val Ile Phe Asp Cys Ser His Glu Met Ala Ala Gly Ile Leu Lys
225 230 235 240
Gln Ala Leu Ala Met Gly Met Met Thr Glu Tyr Tyr His Tyr Ile Phe
245 250 255
Thr Thr Leu Asp Leu Phe Ala Leu Asp Val Glu Pro Tyr Arg Tyr Ser
260 265 270
Gly Val Asn Met Thr Gly Phe Arg Ile Leu Asn Thr Glu Asn Thr Gln
275 280 285
Val Ser Ser Ile Ile Glu Lys Trp Ser Met Glu Arg Leu Gln Ala Pro
290 295 300
Pro Lys Pro Asp Ser Gly Leu Leu Asp Gly Phe Met Thr Thr Asp Ala
305 310 315 320
Ala Leu Met Tyr Asp Ala Val His Val Val Ser Val Ala Val Gln Gln
325 330 335
Phe Pro Gln Met Thr Val Ser Ser Leu Gln Cys Asn Arg His Lys Pro
340 345 350
Trp Arg Phe Gly Thr Arg Phe Met Ser Leu Ile Lys Glu Ala His Trp
355 360 365
Glu Gly Leu Thr Gly Arg Ile Thr Phe Asn Lys Thr Asn Gly Leu Arg
370 375 380
Thr Asp Phe Asp Leu Asp Val Ile Ser Leu Lys Glu Glu Gly Leu Glu
385 390 395 400
Lys Ile Gly Thr Trp Asp Pro Ala Ser Gly Leu Asn Met Thr Glu Ser
405 410 415
Gln Lys Gly Lys Pro Ala Asn Ile Thr Asp Ser Leu Ser Asn Arg Ser
420 425 430
Leu Ile Val Thr Thr Ile Leu Glu Glu Pro Tyr Val Leu Phe Lys Lys
435 440 445
Ser Asp Lys Pro Leu Tyr Gly Asn Asp Arg Phe Glu Gly Tyr Cys Ile
450 455 460
Asp Leu Leu Arg Glu Leu Ser Thr Ile Leu Gly Phe Thr Tyr Glu Ile
465 470 475 480
Arg Leu Val Glu Asp Gly Lys Tyr Gly Ala Gln Asp Asp Ala Asn Gly
485 490 495
Gln Trp Asn Gly Met Val Arg Glu Leu Ile Asp His Lys Ala Asp Leu
500 505 510
Ala Val Ala Pro Leu Ala Ile Thr Tyr Val Arg Glu Lys Val Ile Asp
515 520 525
Phe Ser Lys Pro Phe Met Thr Leu Gly Ile Ser Ile Leu Tyr Arg Lys
530 535 540
Pro Asn Gly Ser Glu Leu Met Pro Lys Ala Leu Ser Thr Arg Ile Val
545 550 555 560
Gly Gly Ile Trp Trp Phe Phe Thr Leu Ile Ile Ile Ser Ser Tyr Thr
565 570 575
Ala Asn Leu Ala Ala Phe Leu Thr Val Glu Arg Met Glu Ser Pro Ile
580 585 590
Asp Ser Ala Asp Asp Leu Ala Lys Gln Thr Lys Ile Glu Tyr Gly Ala
595 600 605
Val Glu Asp Gly Ala Thr Met Thr Phe Phe Lys Lys Ser Lys Ile Ser
610 615 620
Thr Tyr Asp Lys Met Trp Ala Phe Met Ser Ser Arg Arg Gln Ser Val
625 630 635 640
Leu Val Lys Ser Asn Glu Glu Gly Ile Gln Arg Val Leu Thr Ser Asp
645 650 655
Tyr Ala Phe Leu Met Glu Ser Thr Thr Ile Glu Phe Val Thr Gln Arg
660 665 670
Asn Cys Asn Leu Thr Gln Ile Gly Gly Leu Ile Asp Ser Lys Gly Tyr
675 680 685
Gly Val Gly Thr Pro Met Gly Ser Pro Tyr Arg Asp Lys Ile Thr Ile
690 695 700
Ala Ile Leu Gln Leu Gln Glu Glu Gly Lys Leu His Met Met Lys Glu
705 710 715 720
Lys Trp Trp Arg Gly Asn Gly Cys Pro Glu Glu Glu Ser Lys Glu Ala
725 730 735
Ser Ala Leu Gly Val Gln Asn Ile Gly Gly Ile Phe Ile Val Leu Ala
740 745 750
Ala Gly Leu Val Leu Ser Val Phe Val Ala Val Gly Glu Phe Leu Tyr
755 760 765
Lys Ser Lys Lys Asn Ala Gln Leu Glu Lys Arg Ser Phe Cys Ser Ala
770 775 780
Met Val Glu Glu Leu Arg Met Ser Leu Lys Cys Gln Arg Arg Leu Lys
785 790 795 800
His Lys Pro Gln Ala Pro Val Ile Val Lys Thr Glu Glu Val Ile Asn
805 810 815
Met His Thr Phe Asn Asp Arg Arg Leu Pro Gly Lys Glu Thr Met Ala
820 825 830
<210> 106
<211> 892
<212> PRT
<213> Homo sapiens
<400> 106
Met Lys Ile Ile Phe Pro Ile Leu Ser Asn Pro Val Phe Arg Arg Thr
1 5 10 15
Val Lys Leu Leu Leu Cys Leu Leu Trp Ile Gly Tyr Ser Gln Gly Thr
20 25 30
Thr His Val Leu Arg Phe Gly Gly Ile Phe Glu Tyr Val Glu Ser Gly
35 40 45
Pro Met Gly Ala Glu Glu Leu Ala Phe Arg Phe Ala Val Asn Thr Ile
50 55 60
Asn Arg Asn Arg Thr Leu Leu Pro Asn Thr Thr Leu Thr Tyr Asp Thr
65 70 75 80
Gln Lys Ile Asn Leu Tyr Asp Ser Phe Glu Ala Ser Lys Lys Ala Cys
85 90 95
Asp Gln Leu Ser Leu Gly Val Ala Ala Ile Phe Gly Pro Ser His Ser
100 105 110
Ser Ser Ala Asn Ala Val Gln Ser Ile Cys Asn Ala Leu Gly Val Pro
115 120 125
His Ile Gln Thr Arg Trp Lys His Gln Val Ser Asp Asn Lys Asp Ser
130 135 140
Phe Tyr Val Ser Leu Tyr Pro Asp Phe Ser Ser Leu Ser Arg Ala Ile
145 150 155 160
Leu Asp Leu Val Gln Phe Phe Lys Trp Lys Thr Val Thr Val Val Tyr
165 170 175
Asp Asp Ser Thr Gly Leu Ile Arg Leu Gln Glu Leu Ile Lys Ala Pro
180 185 190
Ser Arg Tyr Asn Leu Arg Leu Lys Ile Arg Gln Leu Pro Ala Asp Thr
195 200 205
Lys Asp Ala Lys Pro Leu Leu Lys Glu Met Lys Arg Gly Lys Glu Phe
210 215 220
His Val Ile Phe Asp Cys Ser His Glu Met Ala Ala Gly Ile Leu Lys
225 230 235 240
Gln Ala Leu Ala Met Gly Met Met Thr Glu Tyr Tyr His Tyr Ile Phe
245 250 255
Thr Thr Leu Asp Leu Phe Ala Leu Asp Val Glu Pro Tyr Arg Tyr Ser
260 265 270
Gly Val Asn Met Thr Gly Phe Arg Ile Leu Asn Thr Glu Asn Thr Gln
275 280 285
Val Ser Ser Ile Ile Glu Lys Trp Ser Met Glu Arg Leu Gln Ala Pro
290 295 300
Pro Lys Pro Asp Ser Gly Leu Leu Asp Gly Phe Met Thr Thr Asp Ala
305 310 315 320
Ala Leu Met Tyr Asp Ala Val His Val Val Ser Val Ala Val Gln Gln
325 330 335
Phe Pro Gln Met Thr Val Ser Ser Leu Gln Cys Asn Arg His Lys Pro
340 345 350
Trp Arg Phe Gly Thr Arg Phe Met Ser Leu Ile Lys Glu Ala His Trp
355 360 365
Glu Gly Leu Thr Gly Arg Ile Thr Phe Asn Lys Thr Asn Gly Leu Arg
370 375 380
Thr Asp Phe Asp Leu Asp Val Ile Ser Leu Lys Glu Glu Gly Leu Glu
385 390 395 400
Lys Ile Gly Thr Trp Asp Pro Ala Ser Gly Leu Asn Met Thr Glu Ser
405 410 415
Gln Lys Gly Lys Pro Ala Asn Ile Thr Asp Ser Leu Ser Asn Arg Ser
420 425 430
Leu Ile Val Thr Thr Ile Leu Glu Glu Pro Tyr Val Leu Phe Lys Lys
435 440 445
Ser Asp Lys Pro Leu Tyr Gly Asn Asp Arg Phe Glu Gly Tyr Cys Ile
450 455 460
Asp Leu Leu Arg Glu Leu Ser Thr Ile Leu Gly Phe Thr Tyr Glu Ile
465 470 475 480
Arg Leu Val Glu Asp Gly Lys Tyr Gly Ala Gln Asp Asp Ala Asn Gly
485 490 495
Gln Trp Asn Gly Met Val Arg Glu Leu Ile Asp His Lys Ala Asp Leu
500 505 510
Ala Val Ala Pro Leu Ala Ile Thr Tyr Val Arg Glu Lys Val Ile Asp
515 520 525
Phe Ser Lys Pro Phe Met Thr Leu Gly Ile Ser Ile Leu Tyr Arg Lys
530 535 540
Pro Asn Gly Thr Asn Pro Gly Val Phe Ser Phe Leu Asn Pro Leu Ser
545 550 555 560
Pro Asp Ile Trp Met Tyr Ile Leu Leu Ala Tyr Leu Gly Val Ser Cys
565 570 575
Val Leu Phe Val Ile Ala Arg Phe Ser Pro Tyr Glu Trp Tyr Asn Pro
580 585 590
His Pro Cys Asn Pro Asp Ser Asp Val Val Glu Asn Asn Phe Thr Leu
595 600 605
Leu Asn Ser Phe Trp Phe Gly Val Gly Ala Leu Met Gln Gln Gly Ser
610 615 620
Glu Leu Met Pro Lys Ala Leu Ser Thr Arg Ile Val Gly Gly Ile Trp
625 630 635 640
Trp Phe Phe Thr Leu Ile Ile Ile Ser Ser Tyr Thr Ala Asn Leu Ala
645 650 655
Ala Phe Leu Thr Val Glu Arg Met Glu Ser Pro Ile Asp Ser Ala Asp
660 665 670
Asp Leu Ala Lys Gln Thr Lys Ile Glu Tyr Gly Ala Val Glu Asp Gly
675 680 685
Ala Thr Met Thr Phe Phe Lys Lys Ser Lys Ile Ser Thr Tyr Asp Lys
690 695 700
Met Trp Ala Phe Met Ser Ser Arg Arg Gln Ser Val Leu Val Lys Ser
705 710 715 720
Asn Glu Glu Gly Ile Gln Arg Val Leu Thr Ser Asp Tyr Ala Phe Leu
725 730 735
Met Glu Ser Thr Thr Ile Glu Phe Val Thr Gln Arg Asn Cys Asn Leu
740 745 750
Thr Gln Ile Gly Gly Leu Ile Asp Ser Lys Gly Tyr Gly Val Gly Thr
755 760 765
Pro Met Gly Ser Pro Tyr Arg Asp Lys Ile Thr Ile Ala Ile Leu Gln
770 775 780
Leu Gln Glu Glu Gly Lys Leu His Met Met Lys Glu Lys Trp Trp Arg
785 790 795 800
Gly Asn Gly Cys Pro Glu Glu Glu Ser Lys Glu Ala Ser Ala Leu Gly
805 810 815
Val Gln Asn Ile Gly Gly Ile Phe Ile Val Leu Ala Ala Gly Leu Val
820 825 830
Leu Ser Val Phe Val Ala Val Gly Glu Phe Leu Tyr Lys Ser Lys Lys
835 840 845
Asn Ala Gln Leu Glu Lys Arg Ala Lys Thr Lys Leu Pro Gln Asp Tyr
850 855 860
Val Phe Leu Pro Ile Leu Glu Ser Val Ser Ile Ser Thr Val Leu Ser
865 870 875 880
Ser Ser Pro Ser Ser Ser Ser Leu Ser Ser Cys Ser
885 890
<210> 107
<211> 682
<212> PRT
<213> Homo sapiens
<400> 107
Met Lys Ile Ile Phe Pro Ile Leu Ser Asn Pro Val Phe Arg Arg Thr
1 5 10 15
Val Lys Leu Leu Leu Cys Leu Leu Trp Ile Gly Tyr Ser Gln Gly Thr
20 25 30
Thr His Val Leu Arg Phe Gly Gly Ile Phe Glu Tyr Val Glu Ser Gly
35 40 45
Pro Met Gly Ala Glu Glu Leu Ala Phe Arg Phe Ala Val Asn Thr Ile
50 55 60
Asn Arg Asn Arg Thr Leu Leu Pro Asn Thr Thr Leu Thr Tyr Asp Thr
65 70 75 80
Gln Lys Ile Asn Leu Tyr Asp Ser Phe Glu Ala Ser Lys Lys Ala Cys
85 90 95
Asp Gln Leu Ser Leu Gly Val Ala Ala Ile Phe Gly Pro Ser His Ser
100 105 110
Ser Ser Ala Asn Ala Val Gln Ser Ile Cys Asn Ala Leu Gly Val Pro
115 120 125
His Ile Gln Thr Arg Trp Lys His Gln Val Ser Asp Asn Lys Asp Ser
130 135 140
Phe Tyr Val Ser Leu Tyr Pro Asp Phe Ser Ser Leu Ser Arg Ala Ile
145 150 155 160
Leu Asp Leu Val Gln Phe Phe Lys Trp Lys Thr Val Thr Val Val Tyr
165 170 175
Asp Asp Ser Thr Gly Leu Ile Arg Leu Gln Glu Leu Ile Lys Ala Pro
180 185 190
Ser Arg Tyr Asn Leu Arg Leu Lys Ile Arg Gln Leu Pro Ala Asp Thr
195 200 205
Lys Asp Ala Lys Pro Leu Leu Lys Glu Met Lys Arg Gly Lys Glu Phe
210 215 220
His Val Ile Phe Asp Cys Ser His Glu Met Ala Ala Gly Ile Leu Lys
225 230 235 240
Gln Ala Leu Ala Met Gly Met Met Thr Glu Tyr Tyr His Tyr Ile Phe
245 250 255
Thr Thr Leu Asp Leu Phe Ala Leu Asp Val Glu Pro Tyr Arg Tyr Ser
260 265 270
Gly Val Asn Met Thr Gly Phe Arg Ile Leu Asn Thr Glu Asn Thr Gln
275 280 285
Val Ser Ser Ile Ile Glu Lys Trp Ser Met Glu Arg Leu Gln Ala Pro
290 295 300
Pro Lys Pro Asp Ser Gly Leu Leu Asp Gly Phe Met Thr Thr Asp Ala
305 310 315 320
Ala Leu Met Tyr Asp Ala Val His Val Val Ser Val Ala Val Gln Gln
325 330 335
Phe Pro Gln Met Thr Val Ser Ser Leu Gln Cys Asn Arg His Lys Pro
340 345 350
Trp Arg Phe Gly Thr Arg Phe Met Ser Leu Ile Lys Glu Ala His Trp
355 360 365
Glu Gly Leu Thr Gly Arg Ile Thr Phe Asn Lys Thr Asn Gly Leu Arg
370 375 380
Thr Asp Phe Asp Leu Asp Val Ile Ser Leu Lys Glu Glu Gly Leu Glu
385 390 395 400
Lys Ile Gly Thr Trp Asp Pro Ala Ser Gly Leu Asn Met Thr Glu Ser
405 410 415
Gln Lys Gly Lys Pro Ala Asn Ile Thr Asp Ser Leu Ser Asn Arg Ser
420 425 430
Leu Ile Val Thr Thr Ile Leu Glu Glu Pro Tyr Val Leu Phe Lys Lys
435 440 445
Ser Asp Lys Pro Leu Tyr Gly Asn Asp Arg Phe Glu Gly Tyr Cys Ile
450 455 460
Asp Leu Leu Arg Glu Leu Ser Thr Ile Leu Gly Phe Thr Tyr Glu Ile
465 470 475 480
Arg Leu Val Glu Asp Gly Lys Tyr Gly Ala Gln Asp Asp Ala Asn Gly
485 490 495
Gln Trp Asn Gly Met Val Arg Glu Leu Ile Asp His Lys Ser Lys Ile
500 505 510
Ser Thr Tyr Asp Lys Met Trp Ala Phe Met Ser Ser Arg Arg Gln Ser
515 520 525
Val Leu Val Lys Ser Asn Glu Glu Gly Ile Gln Arg Val Leu Thr Ser
530 535 540
Asp Tyr Ala Phe Leu Met Glu Ser Thr Thr Ile Glu Phe Val Thr Gln
545 550 555 560
Arg Asn Cys Asn Leu Thr Gln Ile Gly Gly Leu Ile Asp Ser Lys Gly
565 570 575
Tyr Gly Val Gly Thr Pro Met Gly Ser Pro Tyr Arg Asp Lys Ile Thr
580 585 590
Ile Ala Ile Leu Gln Leu Gln Glu Glu Gly Lys Leu His Met Met Lys
595 600 605
Glu Lys Trp Trp Arg Gly Asn Gly Cys Pro Glu Glu Glu Ser Lys Glu
610 615 620
Ala Ser Ala Leu Gly Val Gln Asn Ile Gly Gly Ile Phe Ile Val Leu
625 630 635 640
Ala Ala Gly Leu Val Leu Ser Val Phe Val Ala Val Gly Glu Phe Leu
645 650 655
Tyr Lys Ser Lys Lys Asn Ala Gln Leu Glu Lys Glu Ser Ser Ile Trp
660 665 670
Leu Val Pro Pro Tyr His Pro Asp Thr Val
675 680
<210> 108
<211> 687
<212> PRT
<213> Homo sapiens
<400> 108
Met Lys Ile Ile Phe Pro Ile Leu Ser Asn Pro Val Phe Arg Arg Thr
1 5 10 15
Val Lys Leu Leu Leu Cys Leu Leu Trp Ile Gly Tyr Ser Gln Gly Thr
20 25 30
Thr His Val Leu Arg Phe Gly Gly Ile Phe Glu Tyr Val Glu Ser Gly
35 40 45
Pro Met Gly Ala Glu Glu Leu Ala Phe Arg Phe Ala Val Asn Thr Ile
50 55 60
Asn Arg Asn Arg Thr Leu Leu Pro Asn Thr Thr Leu Thr Tyr Asp Thr
65 70 75 80
Gln Lys Ile Asn Leu Tyr Asp Ser Phe Glu Ala Ser Lys Lys Ala Cys
85 90 95
Asp Gln Leu Ser Leu Gly Val Ala Ala Ile Phe Gly Pro Ser His Ser
100 105 110
Ser Ser Ala Asn Ala Val Gln Ser Ile Cys Asn Ala Leu Gly Val Pro
115 120 125
His Ile Gln Thr Arg Trp Lys His Gln Val Ser Asp Asn Lys Asp Ser
130 135 140
Phe Tyr Val Ser Leu Tyr Pro Asp Phe Ser Ser Leu Ser Arg Ala Ile
145 150 155 160
Leu Asp Leu Val Gln Phe Phe Lys Trp Lys Thr Val Thr Val Val Tyr
165 170 175
Asp Asp Ser Thr Gly Leu Ile Arg Leu Gln Glu Leu Ile Lys Ala Pro
180 185 190
Ser Arg Tyr Asn Leu Arg Leu Lys Ile Arg Gln Leu Pro Ala Asp Thr
195 200 205
Lys Asp Ala Lys Pro Leu Leu Lys Glu Met Lys Arg Gly Lys Glu Phe
210 215 220
His Val Ile Phe Asp Cys Ser His Glu Met Ala Ala Gly Ile Leu Lys
225 230 235 240
Gln Ala Leu Ala Met Gly Met Met Thr Glu Tyr Tyr His Tyr Ile Phe
245 250 255
Thr Thr Leu Asp Leu Phe Ala Leu Asp Val Glu Pro Tyr Arg Tyr Ser
260 265 270
Gly Val Asn Met Thr Gly Phe Arg Ile Leu Asn Thr Glu Asn Thr Gln
275 280 285
Val Ser Ser Ile Ile Glu Lys Trp Ser Met Glu Arg Leu Gln Ala Pro
290 295 300
Pro Lys Pro Asp Ser Gly Leu Leu Asp Gly Phe Met Thr Thr Asp Ala
305 310 315 320
Ala Leu Met Tyr Asp Ala Val His Val Val Ser Val Ala Val Gln Gln
325 330 335
Phe Pro Gln Met Thr Val Ser Ser Leu Gln Cys Asn Arg His Lys Pro
340 345 350
Trp Arg Phe Gly Thr Arg Phe Met Ser Leu Ile Lys Glu Ala His Trp
355 360 365
Glu Gly Leu Thr Gly Arg Ile Thr Phe Asn Lys Thr Asn Gly Leu Arg
370 375 380
Thr Asp Phe Asp Leu Asp Val Ile Ser Leu Lys Glu Glu Gly Leu Glu
385 390 395 400
Lys Ile Gly Thr Trp Asp Pro Ala Ser Gly Leu Asn Met Thr Glu Ser
405 410 415
Gln Lys Gly Lys Pro Ala Asn Ile Thr Asp Ser Leu Ser Asn Arg Ser
420 425 430
Leu Ile Val Thr Thr Ile Leu Glu Glu Pro Tyr Val Leu Phe Lys Lys
435 440 445
Ser Asp Lys Pro Leu Tyr Gly Asn Asp Arg Phe Glu Gly Tyr Cys Ile
450 455 460
Asp Leu Leu Arg Glu Leu Ser Thr Ile Leu Gly Phe Thr Tyr Glu Ile
465 470 475 480
Arg Leu Val Glu Asp Gly Lys Tyr Gly Ala Gln Asp Asp Ala Asn Gly
485 490 495
Gln Trp Asn Gly Met Val Arg Glu Leu Ile Asp His Lys Ser Val Leu
500 505 510
Val Lys Ser Asn Glu Glu Gly Ile Gln Arg Val Leu Thr Ser Asp Tyr
515 520 525
Ala Phe Leu Met Glu Ser Thr Thr Ile Glu Phe Val Thr Gln Arg Asn
530 535 540
Cys Asn Leu Thr Gln Ile Gly Gly Leu Ile Asp Ser Lys Gly Tyr Gly
545 550 555 560
Val Gly Thr Pro Met Gly Ser Pro Tyr Arg Asp Lys Ile Thr Ile Ala
565 570 575
Ile Leu Gln Leu Gln Glu Glu Gly Lys Leu His Met Met Lys Glu Lys
580 585 590
Trp Trp Arg Gly Asn Gly Cys Pro Glu Glu Glu Ser Lys Glu Ala Ser
595 600 605
Ala Leu Gly Val Gln Asn Ile Gly Gly Ile Phe Ile Val Leu Ala Ala
610 615 620
Gly Leu Val Leu Ser Val Phe Val Ala Val Gly Glu Phe Leu Tyr Lys
625 630 635 640
Ser Lys Lys Asn Ala Gln Leu Glu Lys Arg Ala Lys Thr Lys Leu Pro
645 650 655
Gln Asp Tyr Val Phe Leu Pro Ile Leu Glu Ser Val Ser Ile Ser Thr
660 665 670
Val Leu Ser Ser Ser Pro Ser Ser Ser Ser Leu Ser Ser Cys Ser
675 680 685
<210> 109
<211> 908
<212> PRT
<213> Mus musculus
<400> 109
Met Lys Ile Ile Ser Pro Val Leu Ser Asn Leu Val Phe Ser Arg Ser
1 5 10 15
Ile Lys Val Leu Leu Cys Leu Leu Trp Ile Gly Tyr Ser Gln Gly Thr
20 25 30
Thr His Val Leu Arg Phe Gly Gly Ile Phe Glu Tyr Val Glu Ser Gly
35 40 45
Pro Met Gly Ala Glu Glu Leu Ala Phe Arg Phe Ala Val Asn Thr Ile
50 55 60
Asn Arg Asn Arg Thr Leu Leu Pro Asn Thr Thr Leu Thr Tyr Asp Thr
65 70 75 80
Gln Lys Ile Asn Leu Tyr Asp Ser Phe Glu Ala Ser Lys Lys Ala Cys
85 90 95
Asp Gln Leu Ser Leu Gly Val Ala Ala Ile Phe Gly Pro Ser His Ser
100 105 110
Ser Ser Ala Asn Ala Val Gln Ser Ile Cys Asn Ala Leu Gly Val Pro
115 120 125
His Ile Gln Thr Arg Trp Lys His Gln Val Ser Asp Asn Lys Asp Ser
130 135 140
Phe Tyr Val Ser Leu Tyr Pro Asp Phe Ser Ser Leu Ser Arg Ala Ile
145 150 155 160
Leu Asp Leu Val Gln Phe Phe Lys Trp Lys Thr Val Thr Val Val Tyr
165 170 175
Asp Asp Ser Thr Gly Leu Ile Arg Leu Gln Glu Leu Ile Lys Ala Pro
180 185 190
Ser Arg Tyr Asn Leu Arg Leu Lys Ile Arg Gln Leu Pro Ala Asp Thr
195 200 205
Lys Asp Ala Lys Pro Leu Leu Lys Glu Met Lys Arg Gly Lys Glu Phe
210 215 220
His Val Ile Phe Asp Cys Ser His Glu Met Ala Ala Gly Ile Leu Lys
225 230 235 240
Gln Ala Leu Ala Met Gly Met Met Thr Glu Tyr Tyr His Tyr Ile Phe
245 250 255
Thr Thr Leu Asp Leu Phe Ala Leu Asp Val Glu Pro Tyr Arg Tyr Ser
260 265 270
Gly Val Asn Met Thr Gly Phe Arg Ile Leu Asn Thr Glu Asn Thr Gln
275 280 285
Val Ser Ser Ile Ile Glu Lys Trp Ser Met Glu Arg Leu Gln Ala Pro
290 295 300
Pro Lys Pro Asp Ser Gly Leu Leu Asp Gly Phe Met Thr Thr Asp Ala
305 310 315 320
Ala Leu Met Tyr Asp Ala Val His Val Val Ser Val Ala Val Gln Gln
325 330 335
Phe Pro Gln Met Thr Val Ser Ser Leu Gln Cys Asn Arg His Lys Pro
340 345 350
Trp Arg Phe Gly Thr Arg Phe Met Ser Leu Ile Lys Glu Ala His Trp
355 360 365
Glu Gly Leu Thr Gly Arg Ile Thr Phe Asn Lys Thr Asn Gly Leu Arg
370 375 380
Thr Asp Phe Asp Leu Asp Val Ile Ser Leu Lys Glu Glu Gly Leu Glu
385 390 395 400
Lys Ile Gly Thr Trp Asp Pro Ser Ser Gly Leu Asn Met Thr Glu Ser
405 410 415
Gln Lys Gly Lys Pro Ala Asn Ile Thr Asp Ser Leu Ser Asn Arg Ser
420 425 430
Leu Ile Val Thr Thr Ile Leu Glu Glu Pro Tyr Val Leu Phe Lys Lys
435 440 445
Ser Asp Lys Pro Leu Tyr Gly Asn Asp Arg Phe Glu Gly Tyr Cys Ile
450 455 460
Asp Leu Leu Arg Glu Leu Ser Thr Ile Leu Gly Phe Thr Tyr Glu Ile
465 470 475 480
Arg Leu Val Glu Asp Gly Lys Tyr Gly Ala Gln Asp Asp Val Asn Gly
485 490 495
Gln Trp Asn Gly Met Val Arg Glu Leu Ile Asp His Lys Ala Asp Leu
500 505 510
Ala Val Ala Pro Leu Ala Ile Thr Tyr Val Arg Glu Lys Val Ile Asp
515 520 525
Phe Ser Lys Pro Phe Met Thr Leu Gly Ile Ser Ile Leu Tyr Arg Lys
530 535 540
Pro Asn Gly Thr Asn Pro Gly Val Phe Ser Phe Leu Asn Pro Leu Ser
545 550 555 560
Pro Asp Ile Trp Met Tyr Ile Leu Leu Ala Tyr Leu Gly Val Ser Cys
565 570 575
Val Leu Phe Val Ile Ala Arg Phe Ser Pro Tyr Glu Trp Tyr Asn Pro
580 585 590
His Pro Cys Asn Pro Asp Ser Asp Val Val Glu Asn Asn Phe Thr Leu
595 600 605
Leu Asn Ser Phe Trp Phe Gly Val Gly Ala Leu Met Gln Gln Gly Ser
610 615 620
Glu Leu Met Pro Lys Ala Leu Ser Thr Arg Ile Val Gly Gly Ile Trp
625 630 635 640
Trp Phe Phe Thr Leu Ile Ile Ile Ser Ser Tyr Thr Ala Asn Leu Ala
645 650 655
Ala Phe Leu Thr Val Glu Arg Met Glu Ser Pro Ile Asp Ser Ala Asp
660 665 670
Asp Leu Ala Lys Gln Thr Lys Ile Glu Tyr Gly Ala Val Glu Asp Gly
675 680 685
Ala Thr Met Thr Phe Phe Lys Lys Ser Lys Ile Ser Thr Tyr Asp Lys
690 695 700
Met Trp Ala Phe Met Ser Ser Arg Arg Gln Ser Val Leu Val Lys Ser
705 710 715 720
Asn Glu Glu Gly Ile Gln Arg Val Leu Thr Ser Asp Tyr Ala Phe Leu
725 730 735
Met Glu Ser Thr Thr Ile Glu Phe Val Thr Gln Arg Asn Cys Asn Leu
740 745 750
Thr Gln Ile Gly Gly Leu Ile Asp Ser Lys Gly Tyr Gly Val Gly Thr
755 760 765
Pro Met Gly Ser Pro Tyr Arg Asp Lys Ile Thr Ile Ala Ile Leu Gln
770 775 780
Leu Gln Glu Glu Gly Lys Leu His Met Met Lys Glu Lys Trp Trp Arg
785 790 795 800
Gly Asn Gly Cys Pro Glu Glu Glu Ser Lys Glu Ala Ser Ala Leu Gly
805 810 815
Val Gln Asn Ile Gly Gly Ile Phe Ile Val Leu Ala Ala Gly Leu Val
820 825 830
Leu Ser Val Phe Val Ala Val Gly Glu Phe Leu Tyr Lys Ser Lys Lys
835 840 845
Asn Ala Gln Leu Glu Lys Arg Ser Phe Cys Ser Ala Met Val Glu Glu
850 855 860
Leu Arg Met Ser Leu Lys Cys Gln Arg Arg Leu Lys His Lys Pro Gln
865 870 875 880
Ala Pro Val Ile Val Lys Thr Glu Glu Val Ile Asn Met His Thr Phe
885 890 895
Asn Asp Arg Arg Leu Pro Gly Lys Glu Thr Met Ala
900 905
<210> 110
<211> 869
<212> PRT
<213> Mus musculus
<400> 110
Met Lys Ile Ile Ser Pro Val Leu Ser Asn Leu Val Phe Ser Arg Ser
1 5 10 15
Ile Lys Val Leu Leu Cys Leu Leu Trp Ile Gly Tyr Ser Gln Gly Thr
20 25 30
Thr His Val Leu Arg Phe Gly Gly Ile Phe Glu Tyr Val Glu Ser Gly
35 40 45
Pro Met Gly Ala Glu Glu Leu Ala Phe Arg Phe Ala Val Asn Thr Ile
50 55 60
Asn Arg Asn Arg Thr Leu Leu Pro Asn Thr Thr Leu Thr Tyr Asp Thr
65 70 75 80
Gln Lys Ile Asn Leu Tyr Asp Ser Phe Glu Ala Ser Lys Lys Ala Cys
85 90 95
Asp Gln Leu Ser Leu Gly Val Ala Ala Ile Phe Gly Pro Ser His Ser
100 105 110
Ser Ser Ala Asn Ala Val Gln Ser Ile Cys Asn Ala Leu Gly Val Pro
115 120 125
His Ile Gln Thr Arg Trp Lys His Gln Val Ser Asp Asn Lys Asp Ser
130 135 140
Phe Tyr Val Ser Leu Tyr Pro Asp Phe Ser Ser Leu Ser Arg Ala Ile
145 150 155 160
Leu Asp Leu Val Gln Phe Phe Lys Trp Lys Thr Val Thr Val Val Tyr
165 170 175
Asp Asp Ser Thr Gly Leu Ile Arg Leu Gln Glu Leu Ile Lys Ala Pro
180 185 190
Ser Arg Tyr Asn Leu Arg Leu Lys Ile Arg Gln Leu Pro Ala Asp Thr
195 200 205
Lys Asp Ala Lys Pro Leu Leu Lys Glu Met Lys Arg Gly Lys Glu Phe
210 215 220
His Val Ile Phe Asp Cys Ser His Glu Met Ala Ala Gly Ile Leu Lys
225 230 235 240
Gln Ala Leu Ala Met Gly Met Met Thr Glu Tyr Tyr His Tyr Ile Phe
245 250 255
Thr Thr Leu Asp Leu Phe Ala Leu Asp Val Glu Pro Tyr Arg Tyr Ser
260 265 270
Gly Val Asn Met Thr Gly Phe Arg Ile Leu Asn Thr Glu Asn Thr Gln
275 280 285
Val Ser Ser Ile Ile Glu Lys Trp Ser Met Glu Arg Leu Gln Ala Pro
290 295 300
Pro Lys Pro Asp Ser Gly Leu Leu Asp Gly Phe Met Thr Thr Asp Ala
305 310 315 320
Ala Leu Met Tyr Asp Ala Val His Val Val Ser Val Ala Val Gln Gln
325 330 335
Phe Pro Gln Met Thr Val Ser Ser Leu Gln Cys Asn Arg His Lys Pro
340 345 350
Trp Arg Phe Gly Thr Arg Phe Met Ser Leu Ile Lys Glu Ala His Trp
355 360 365
Glu Gly Leu Thr Gly Arg Ile Thr Phe Asn Lys Thr Asn Gly Leu Arg
370 375 380
Thr Asp Phe Asp Leu Asp Val Ile Ser Leu Lys Glu Glu Gly Leu Glu
385 390 395 400
Lys Ile Gly Thr Trp Asp Pro Ser Ser Gly Leu Asn Met Thr Glu Ser
405 410 415
Gln Lys Gly Lys Pro Ala Asn Ile Thr Asp Ser Leu Ser Asn Arg Ser
420 425 430
Leu Ile Val Thr Thr Ile Leu Glu Glu Pro Tyr Val Leu Phe Lys Lys
435 440 445
Ser Asp Lys Pro Leu Tyr Gly Asn Asp Arg Phe Glu Gly Tyr Cys Ile
450 455 460
Asp Leu Leu Arg Glu Leu Ser Thr Ile Leu Gly Phe Thr Tyr Glu Ile
465 470 475 480
Arg Leu Val Glu Asp Gly Lys Tyr Gly Ala Gln Asp Asp Val Asn Gly
485 490 495
Gln Trp Asn Gly Met Val Arg Glu Leu Ile Asp His Lys Ala Asp Leu
500 505 510
Ala Val Ala Pro Leu Ala Ile Thr Tyr Val Arg Glu Lys Val Ile Asp
515 520 525
Phe Ser Lys Pro Phe Met Thr Leu Gly Ile Ser Ile Leu Tyr Arg Lys
530 535 540
Pro Asn Gly Thr Asn Pro Gly Val Phe Ser Phe Leu Asn Pro Leu Ser
545 550 555 560
Pro Asp Ile Trp Met Tyr Ile Leu Leu Ala Tyr Leu Gly Val Ser Cys
565 570 575
Val Leu Phe Val Ile Ala Arg Phe Ser Pro Tyr Glu Trp Tyr Asn Pro
580 585 590
His Pro Cys Asn Pro Asp Ser Asp Val Val Glu Asn Asn Phe Thr Leu
595 600 605
Leu Asn Ser Phe Trp Phe Gly Val Gly Ala Leu Met Gln Gln Gly Ser
610 615 620
Glu Leu Met Pro Lys Ala Leu Ser Thr Arg Ile Val Gly Gly Ile Trp
625 630 635 640
Trp Phe Phe Thr Leu Ile Ile Ile Ser Ser Tyr Thr Ala Asn Leu Ala
645 650 655
Ala Phe Leu Thr Val Glu Arg Met Glu Ser Pro Ile Asp Ser Ala Asp
660 665 670
Asp Leu Ala Lys Gln Thr Lys Ile Glu Tyr Gly Ala Val Glu Asp Gly
675 680 685
Ala Thr Met Thr Phe Phe Lys Lys Ser Lys Ile Ser Thr Tyr Asp Lys
690 695 700
Met Trp Ala Phe Met Ser Ser Arg Arg Gln Ser Val Leu Val Lys Ser
705 710 715 720
Asn Glu Glu Gly Ile Gln Arg Val Leu Thr Ser Asp Tyr Ala Phe Leu
725 730 735
Met Glu Ser Thr Thr Ile Glu Phe Val Thr Gln Arg Asn Cys Asn Leu
740 745 750
Thr Gln Ile Gly Gly Leu Ile Asp Ser Lys Gly Tyr Gly Val Gly Thr
755 760 765
Pro Met Gly Ser Pro Tyr Arg Asp Lys Ile Thr Ile Ala Ile Leu Gln
770 775 780
Leu Gln Glu Glu Gly Lys Leu His Met Met Lys Glu Lys Trp Trp Arg
785 790 795 800
Gly Asn Gly Cys Pro Glu Glu Glu Ser Lys Glu Ala Ser Ala Leu Gly
805 810 815
Val Gln Asn Ile Gly Gly Ile Phe Ile Val Leu Ala Ala Gly Leu Val
820 825 830
Leu Ser Val Phe Val Ala Val Gly Glu Phe Leu Tyr Lys Ser Lys Lys
835 840 845
Asn Ala Gln Leu Glu Lys Glu Ser Ser Ile Trp Leu Val Pro Pro Tyr
850 855 860
His Pro Asp Thr Val
865
<210> 111
<211> 908
<212> PRT
<213> Mus musculus
<400> 111
Met Lys Ile Ile Ser Pro Val Leu Ser Asn Leu Val Phe Ser Arg Ser
1 5 10 15
Ile Lys Val Leu Leu Cys Leu Leu Trp Ile Gly Tyr Ser Gln Gly Thr
20 25 30
Thr His Val Leu Arg Phe Gly Gly Ile Phe Glu Tyr Val Glu Ser Gly
35 40 45
Pro Met Gly Ala Glu Glu Leu Ala Phe Arg Phe Ala Val Asn Thr Ile
50 55 60
Asn Arg Asn Arg Thr Leu Leu Pro Asn Thr Thr Leu Thr Tyr Asp Thr
65 70 75 80
Gln Lys Ile Asn Leu Tyr Asp Ser Phe Glu Ala Ser Lys Lys Ala Cys
85 90 95
Asp Gln Leu Ser Leu Gly Val Ala Ala Ile Phe Gly Pro Ser His Ser
100 105 110
Ser Ser Ala Asn Ala Val Gln Ser Ile Cys Asn Ala Leu Gly Val Pro
115 120 125
His Ile Gln Thr Arg Trp Lys His Gln Val Ser Asp Asn Lys Asp Ser
130 135 140
Phe Tyr Val Ser Leu Tyr Pro Asp Phe Ser Ser Leu Ser Arg Ala Ile
145 150 155 160
Leu Asp Leu Val Gln Phe Phe Lys Trp Lys Thr Val Thr Val Val Tyr
165 170 175
Asp Asp Ser Thr Gly Leu Ile Arg Leu Gln Glu Leu Ile Lys Ala Pro
180 185 190
Ser Arg Tyr Asn Leu Arg Leu Lys Ile Arg Gln Leu Pro Ala Asp Thr
195 200 205
Lys Asp Ala Lys Pro Leu Leu Lys Glu Met Lys Arg Gly Lys Glu Phe
210 215 220
His Val Ile Phe Asp Cys Ser His Glu Met Ala Ala Gly Ile Leu Lys
225 230 235 240
Gln Ala Leu Ala Met Gly Met Met Thr Glu Tyr Tyr His Tyr Ile Phe
245 250 255
Thr Thr Leu Asp Leu Phe Ala Leu Asp Val Glu Pro Tyr Arg Tyr Ser
260 265 270
Gly Val Asn Met Thr Gly Phe Arg Ile Leu Asn Thr Glu Asn Thr Gln
275 280 285
Val Ser Ser Ile Ile Glu Lys Trp Ser Met Glu Arg Leu Gln Ala Pro
290 295 300
Pro Lys Pro Asp Ser Gly Leu Leu Asp Gly Phe Met Thr Thr Asp Ala
305 310 315 320
Ala Leu Met Tyr Asp Ala Val His Val Val Ser Val Ala Val Gln Gln
325 330 335
Phe Pro Gln Met Thr Val Ser Ser Leu Gln Cys Asn Arg His Lys Pro
340 345 350
Trp Arg Phe Gly Thr Arg Phe Met Ser Leu Ile Lys Glu Ala His Trp
355 360 365
Glu Gly Leu Thr Gly Arg Ile Thr Phe Asn Lys Thr Asn Gly Leu Arg
370 375 380
Thr Asp Phe Asp Leu Asp Val Ile Ser Leu Lys Glu Glu Gly Leu Glu
385 390 395 400
Lys Ile Gly Thr Trp Asp Pro Ser Ser Gly Leu Asn Met Thr Glu Ser
405 410 415
Gln Lys Gly Lys Pro Ala Asn Ile Thr Asp Ser Leu Ser Asn Arg Ser
420 425 430
Leu Ile Val Thr Thr Ile Leu Glu Glu Pro Tyr Val Leu Phe Lys Lys
435 440 445
Ser Asp Lys Pro Leu Tyr Gly Asn Asp Arg Phe Glu Gly Tyr Cys Ile
450 455 460
Asp Leu Leu Arg Glu Leu Ser Thr Ile Leu Gly Phe Thr Tyr Glu Ile
465 470 475 480
Arg Leu Val Glu Asp Gly Lys Tyr Gly Ala Gln Asp Asp Val Asn Gly
485 490 495
Gln Trp Asn Gly Met Val Arg Glu Leu Ile Asp His Lys Ala Asp Leu
500 505 510
Ala Val Ala Pro Leu Ala Ile Thr Tyr Val Arg Glu Lys Val Ile Asp
515 520 525
Phe Ser Lys Pro Phe Met Thr Leu Gly Ile Ser Ile Leu Tyr Arg Lys
530 535 540
Pro Asn Gly Thr Asn Pro Gly Val Phe Ser Phe Leu Asn Pro Leu Ser
545 550 555 560
Pro Asp Ile Trp Met Tyr Ile Leu Leu Ala Tyr Leu Gly Val Ser Cys
565 570 575
Val Leu Phe Val Ile Ala Arg Phe Ser Pro Tyr Glu Trp Tyr Asn Pro
580 585 590
His Pro Cys Asn Pro Asp Ser Asp Val Val Glu Asn Asn Phe Thr Leu
595 600 605
Leu Asn Ser Phe Trp Phe Gly Val Gly Ala Leu Met Gln Gln Gly Ser
610 615 620
Glu Leu Met Pro Lys Ala Leu Ser Thr Arg Ile Val Gly Gly Ile Trp
625 630 635 640
Trp Phe Phe Thr Leu Ile Ile Ile Ser Ser Tyr Thr Ala Asn Leu Ala
645 650 655
Ala Phe Leu Thr Val Glu Arg Met Glu Ser Pro Ile Asp Ser Ala Asp
660 665 670
Asp Leu Ala Lys Gln Thr Lys Ile Glu Tyr Gly Ala Val Glu Asp Gly
675 680 685
Ala Thr Met Thr Phe Phe Lys Lys Ser Lys Ile Ser Thr Tyr Asp Lys
690 695 700
Met Trp Ala Phe Met Ser Ser Arg Arg Gln Ser Val Leu Val Lys Ser
705 710 715 720
Asn Glu Glu Gly Ile Gln Arg Val Leu Thr Ser Asp Tyr Ala Phe Leu
725 730 735
Met Glu Ser Thr Thr Ile Glu Phe Val Thr Gln Arg Asn Cys Asn Leu
740 745 750
Thr Gln Ile Gly Gly Leu Ile Asp Ser Lys Gly Tyr Gly Val Gly Thr
755 760 765
Pro Met Gly Ser Pro Tyr Arg Asp Lys Ile Thr Ile Ala Ile Leu Gln
770 775 780
Leu Gln Glu Glu Gly Lys Leu His Met Met Lys Glu Lys Trp Trp Arg
785 790 795 800
Gly Asn Gly Cys Pro Glu Glu Glu Ser Lys Glu Ala Ser Ala Leu Gly
805 810 815
Val Gln Asn Ile Gly Gly Ile Phe Ile Val Leu Ala Ala Gly Leu Val
820 825 830
Leu Ser Val Phe Val Ala Val Gly Glu Phe Leu Tyr Lys Ser Lys Lys
835 840 845
Asn Ala Gln Leu Glu Lys Arg Ser Phe Cys Ser Ala Met Val Glu Glu
850 855 860
Leu Arg Met Ser Leu Lys Cys Gln Arg Arg Leu Lys His Lys Pro Gln
865 870 875 880
Ala Pro Val Ile Val Lys Thr Glu Glu Val Ile Asn Met His Thr Phe
885 890 895
Asn Asp Arg Arg Leu Pro Gly Lys Glu Thr Met Ala
900 905
<210> 112
<211> 908
<212> PRT
<213> Macaca mulatta
<400> 112
Met Lys Ile Ile Phe Pro Ile Leu Ser Asn Pro Val Phe Arg Arg Thr
1 5 10 15
Val Lys Leu Leu Leu Cys Leu Leu Trp Ile Gly Tyr Ser Gln Gly Thr
20 25 30
Thr His Val Leu Arg Phe Gly Gly Ile Phe Glu Tyr Val Glu Ser Gly
35 40 45
Pro Met Gly Ala Glu Glu Leu Ala Phe Arg Phe Ala Val Asn Thr Ile
50 55 60
Asn Arg Asn Arg Thr Leu Leu Pro Asn Thr Thr Leu Thr Tyr Asp Thr
65 70 75 80
Gln Lys Ile Asn Leu Tyr Asp Ser Phe Glu Ala Ser Lys Lys Ala Cys
85 90 95
Asp Gln Leu Ser Leu Gly Val Ala Ala Ile Phe Gly Pro Ser His Ser
100 105 110
Ser Ser Ala Asn Ala Val Gln Ser Ile Cys Asn Ala Leu Gly Val Pro
115 120 125
His Ile Gln Thr Arg Trp Lys His Gln Val Ser Asp Asn Lys Asp Ser
130 135 140
Phe Tyr Val Ser Leu Tyr Pro Asp Phe Ser Ser Leu Ser Arg Ala Ile
145 150 155 160
Leu Asp Leu Val Gln Phe Phe Lys Trp Lys Thr Val Thr Val Val Tyr
165 170 175
Asp Asp Ser Thr Gly Leu Ile Arg Leu Gln Glu Leu Ile Lys Ala Pro
180 185 190
Ser Arg Tyr Asn Leu Arg Leu Lys Ile Arg Gln Leu Pro Ala Asp Thr
195 200 205
Lys Asp Ala Lys Pro Leu Leu Lys Glu Met Lys Arg Gly Lys Glu Phe
210 215 220
His Val Ile Phe Asp Cys Ser His Glu Met Ala Ala Gly Ile Leu Lys
225 230 235 240
Gln Ala Leu Ala Met Gly Met Met Thr Glu Tyr Tyr His Tyr Ile Phe
245 250 255
Thr Thr Leu Asp Leu Phe Ala Leu Asp Val Glu Pro Tyr Arg Tyr Ser
260 265 270
Gly Val Asn Met Thr Gly Phe Arg Ile Leu Asn Thr Glu Asn Thr Gln
275 280 285
Val Ser Ser Ile Ile Glu Lys Trp Ser Met Glu Arg Leu Gln Ala Pro
290 295 300
Pro Lys Pro Asp Ser Gly Leu Leu Asp Gly Phe Met Thr Thr Asp Ala
305 310 315 320
Ala Leu Met Tyr Asp Ala Val His Val Val Ser Val Ala Val Gln Gln
325 330 335
Phe Pro Gln Met Thr Val Ser Ser Leu Gln Cys Asn Arg His Lys Pro
340 345 350
Trp Arg Phe Gly Thr Arg Phe Met Ser Leu Ile Lys Glu Ala His Trp
355 360 365
Glu Gly Leu Thr Gly Arg Ile Thr Phe Asn Lys Thr Asn Gly Leu Arg
370 375 380
Thr Asp Phe Asp Leu Asp Val Ile Ser Leu Lys Glu Glu Gly Leu Glu
385 390 395 400
Lys Ile Gly Thr Trp Asp Pro Ala Ser Gly Leu Asn Met Thr Glu Ser
405 410 415
Gln Lys Gly Lys Pro Ala Asn Ile Thr Asp Ser Leu Ser Asn Arg Ser
420 425 430
Leu Ile Val Thr Thr Ile Leu Glu Glu Pro Tyr Val Leu Phe Lys Lys
435 440 445
Ser Asp Lys Pro Leu Tyr Gly Asn Asp Arg Phe Glu Gly Tyr Cys Ile
450 455 460
Asp Leu Leu Arg Glu Leu Ser Thr Ile Leu Gly Phe Thr Tyr Glu Ile
465 470 475 480
Arg Leu Val Glu Asp Gly Lys Tyr Gly Ala Gln Asp Asp Ala Asn Gly
485 490 495
Gln Trp Asn Gly Met Val Arg Glu Leu Ile Asp His Lys Ala Asp Leu
500 505 510
Ala Val Ala Pro Leu Ala Ile Thr Tyr Val Arg Glu Lys Val Ile Asp
515 520 525
Phe Ser Lys Pro Phe Met Thr Leu Gly Ile Ser Ile Leu Tyr Arg Lys
530 535 540
Pro Asn Gly Thr Asn Pro Gly Val Phe Ser Phe Leu Asn Pro Leu Ser
545 550 555 560
Pro Asp Ile Trp Met Tyr Ile Leu Leu Ala Tyr Leu Gly Val Ser Cys
565 570 575
Val Leu Phe Val Ile Ala Arg Phe Ser Pro Tyr Glu Trp Tyr Asn Pro
580 585 590
His Pro Cys Asn Pro Asp Ser Asp Val Val Glu Asn Asn Phe Thr Leu
595 600 605
Leu Asn Ser Phe Trp Phe Gly Val Gly Ala Leu Met Gln Gln Gly Ser
610 615 620
Glu Leu Met Pro Lys Ala Leu Ser Thr Arg Ile Val Gly Gly Ile Trp
625 630 635 640
Trp Phe Phe Thr Leu Ile Ile Ile Ser Ser Tyr Thr Ala Asn Leu Ala
645 650 655
Ala Phe Leu Thr Val Glu Arg Met Glu Ser Pro Ile Asp Ser Ala Asp
660 665 670
Asp Leu Ala Lys Gln Thr Lys Ile Glu Tyr Gly Ala Val Glu Asp Gly
675 680 685
Ala Thr Met Thr Phe Phe Lys Lys Ser Lys Ile Ser Thr Tyr Asp Lys
690 695 700
Met Trp Ala Phe Met Ser Ser Arg Arg Gln Ser Val Leu Val Lys Ser
705 710 715 720
Asn Glu Glu Gly Ile Gln Arg Val Leu Thr Ser Asp Tyr Ala Phe Leu
725 730 735
Met Glu Ser Thr Thr Ile Glu Phe Val Thr Gln Arg Asn Cys Asn Leu
740 745 750
Thr Gln Ile Gly Gly Leu Ile Asp Ser Lys Gly Tyr Gly Val Gly Thr
755 760 765
Pro Met Gly Ser Pro Tyr Arg Asp Lys Ile Thr Ile Ala Ile Leu Gln
770 775 780
Leu Gln Glu Glu Gly Lys Leu His Met Met Lys Glu Lys Trp Trp Arg
785 790 795 800
Gly Asn Gly Cys Pro Glu Glu Glu Ser Lys Glu Ala Ser Ala Leu Gly
805 810 815
Val Gln Asn Ile Gly Gly Ile Phe Ile Val Leu Ala Ala Gly Leu Val
820 825 830
Leu Ser Val Phe Val Ala Val Gly Glu Phe Leu Tyr Lys Ser Lys Lys
835 840 845
Asn Ala Gln Leu Glu Lys Arg Ser Phe Cys Ser Ala Met Val Glu Glu
850 855 860
Leu Arg Met Ser Leu Lys Cys Gln Arg Arg Leu Lys His Lys Pro Gln
865 870 875 880
Ala Pro Val Ile Val Lys Thr Glu Glu Val Ile Asn Met His Thr Phe
885 890 895
Asn Asp Arg Arg Leu Pro Gly Lys Glu Thr Met Ala
900 905
<210> 113
<211> 908
<212> PRT
<213> Macaca mulatta
<400> 113
Met Lys Ile Ile Phe Pro Ile Leu Ser Asn Pro Val Phe Arg Arg Thr
1 5 10 15
Val Lys Leu Leu Leu Cys Leu Leu Trp Ile Gly Tyr Ser Gln Gly Thr
20 25 30
Thr His Val Leu Arg Phe Gly Gly Ile Phe Glu Tyr Val Glu Ser Gly
35 40 45
Pro Met Gly Ala Glu Glu Leu Ala Phe Arg Phe Ala Val Asn Thr Ile
50 55 60
Asn Arg Asn Arg Thr Leu Leu Pro Asn Thr Thr Leu Thr Tyr Asp Thr
65 70 75 80
Gln Lys Ile Asn Leu Tyr Asp Ser Phe Glu Ala Ser Lys Lys Ala Cys
85 90 95
Asp Gln Leu Ser Leu Gly Val Ala Ala Ile Phe Gly Pro Ser His Ser
100 105 110
Ser Ser Ala Asn Ala Val Gln Ser Ile Cys Asn Ala Leu Gly Val Pro
115 120 125
His Ile Gln Thr Arg Trp Lys His Gln Val Ser Asp Asn Lys Asp Ser
130 135 140
Phe Tyr Val Ser Leu Tyr Pro Asp Phe Ser Ser Leu Ser Arg Ala Ile
145 150 155 160
Leu Asp Leu Val Gln Phe Phe Lys Trp Lys Thr Val Thr Val Val Tyr
165 170 175
Asp Asp Ser Thr Gly Leu Ile Arg Leu Gln Glu Leu Ile Lys Ala Pro
180 185 190
Ser Arg Tyr Asn Leu Arg Leu Lys Ile Arg Gln Leu Pro Ala Asp Thr
195 200 205
Lys Asp Ala Lys Pro Leu Leu Lys Glu Met Lys Arg Gly Lys Glu Phe
210 215 220
His Val Ile Phe Asp Cys Ser His Glu Met Ala Ala Gly Ile Leu Lys
225 230 235 240
Gln Ala Leu Ala Met Gly Met Met Thr Glu Tyr Tyr His Tyr Ile Phe
245 250 255
Thr Thr Leu Asp Leu Phe Ala Leu Asp Val Glu Pro Tyr Arg Tyr Ser
260 265 270
Gly Val Asn Met Thr Gly Phe Arg Ile Leu Asn Thr Glu Asn Thr Gln
275 280 285
Val Ser Ser Ile Ile Glu Lys Trp Ser Met Glu Arg Leu Gln Ala Pro
290 295 300
Pro Lys Pro Asp Ser Gly Leu Leu Asp Gly Phe Met Thr Thr Asp Ala
305 310 315 320
Ala Leu Met Tyr Asp Ala Val His Val Val Ser Val Ala Val Gln Gln
325 330 335
Phe Pro Gln Met Thr Val Ser Ser Leu Gln Cys Asn Arg His Lys Pro
340 345 350
Trp Arg Phe Gly Thr Arg Phe Met Ser Leu Ile Lys Glu Ala His Trp
355 360 365
Glu Gly Leu Thr Gly Arg Ile Thr Phe Asn Lys Thr Asn Gly Leu Arg
370 375 380
Thr Asp Phe Asp Leu Asp Val Ile Ser Leu Lys Glu Glu Gly Leu Glu
385 390 395 400
Lys Ile Gly Thr Trp Asp Pro Ala Ser Gly Leu Asn Met Thr Glu Ser
405 410 415
Gln Lys Gly Lys Pro Ala Asn Ile Thr Asp Ser Leu Ser Asn Arg Ser
420 425 430
Leu Ile Val Thr Thr Ile Leu Glu Glu Pro Tyr Val Leu Phe Lys Lys
435 440 445
Ser Asp Lys Pro Leu Tyr Gly Asn Asp Arg Phe Glu Gly Tyr Cys Ile
450 455 460
Asp Leu Leu Arg Glu Leu Ser Thr Ile Leu Gly Phe Thr Tyr Glu Ile
465 470 475 480
Arg Leu Val Glu Asp Gly Lys Tyr Gly Ala Gln Asp Asp Ala Asn Gly
485 490 495
Gln Trp Asn Gly Met Val Arg Glu Leu Ile Asp His Lys Ala Asp Leu
500 505 510
Ala Val Ala Pro Leu Ala Ile Thr Tyr Val Arg Glu Lys Val Ile Asp
515 520 525
Phe Ser Lys Pro Phe Met Thr Leu Gly Ile Ser Ile Leu Tyr Arg Lys
530 535 540
Pro Asn Gly Thr Asn Pro Gly Val Phe Ser Phe Leu Asn Pro Leu Ser
545 550 555 560
Pro Asp Ile Trp Met Tyr Ile Leu Leu Ala Tyr Leu Gly Val Ser Cys
565 570 575
Val Leu Phe Val Ile Ala Arg Phe Ser Pro Tyr Glu Trp Tyr Asn Pro
580 585 590
His Pro Cys Asn Pro Asp Ser Asp Val Val Glu Asn Asn Phe Thr Leu
595 600 605
Leu Asn Ser Phe Trp Phe Gly Val Gly Ala Leu Met Gln Gln Gly Ser
610 615 620
Glu Leu Met Pro Lys Ala Leu Ser Thr Arg Ile Val Gly Gly Ile Trp
625 630 635 640
Trp Phe Phe Thr Leu Ile Ile Ile Ser Ser Tyr Thr Ala Asn Leu Ala
645 650 655
Ala Phe Leu Thr Val Glu Arg Met Glu Ser Pro Ile Asp Ser Ala Asp
660 665 670
Asp Leu Ala Lys Gln Thr Lys Ile Glu Tyr Gly Ala Val Glu Asp Gly
675 680 685
Ala Thr Met Thr Phe Phe Lys Lys Ser Lys Ile Ser Thr Tyr Asp Lys
690 695 700
Met Trp Ala Phe Met Ser Ser Arg Arg Gln Ser Val Leu Val Lys Ser
705 710 715 720
Asn Glu Glu Gly Ile Gln Arg Val Leu Thr Ser Asp Tyr Ala Phe Leu
725 730 735
Met Glu Ser Thr Thr Ile Glu Phe Val Thr Gln Arg Asn Cys Asn Leu
740 745 750
Thr Gln Ile Gly Gly Leu Ile Asp Ser Lys Gly Tyr Gly Val Gly Thr
755 760 765
Pro Met Gly Ser Pro Tyr Arg Asp Lys Ile Thr Ile Ala Ile Leu Gln
770 775 780
Leu Gln Glu Glu Gly Lys Leu His Met Met Lys Glu Lys Trp Trp Arg
785 790 795 800
Gly Asn Gly Cys Pro Glu Glu Glu Ser Lys Glu Ala Ser Ala Leu Gly
805 810 815
Val Gln Asn Ile Gly Gly Ile Phe Ile Val Leu Ala Ala Gly Leu Val
820 825 830
Leu Ser Val Phe Val Ala Val Gly Glu Phe Leu Tyr Lys Ser Lys Lys
835 840 845
Asn Ala Gln Leu Glu Lys Arg Ser Phe Cys Ser Ala Met Val Glu Glu
850 855 860
Leu Arg Met Ser Leu Lys Cys Gln Arg Arg Leu Lys His Lys Pro Gln
865 870 875 880
Ala Pro Val Ile Val Lys Thr Glu Glu Val Ile Asn Met His Thr Phe
885 890 895
Asn Asp Arg Arg Leu Pro Gly Lys Glu Thr Met Ala
900 905
<210> 114
<211> 908
<212> PRT
<213> Rattus norvegicus
<400> 114
Met Lys Ile Ile Ser Pro Val Leu Ser Asn Leu Val Phe Ser Arg Ser
1 5 10 15
Ile Lys Val Leu Leu Cys Leu Leu Trp Ile Gly Tyr Ser Gln Gly Thr
20 25 30
Thr His Val Leu Arg Phe Gly Gly Ile Phe Glu Tyr Val Glu Ser Gly
35 40 45
Pro Met Gly Ala Glu Glu Leu Ala Phe Arg Phe Ala Val Asn Thr Ile
50 55 60
Asn Arg Asn Arg Thr Leu Leu Pro Asn Thr Thr Leu Thr Tyr Asp Thr
65 70 75 80
Gln Lys Ile Asn Leu Tyr Asp Ser Phe Glu Ala Ser Lys Lys Ala Cys
85 90 95
Asp Gln Leu Ser Leu Gly Val Ala Ala Ile Phe Gly Pro Ser His Ser
100 105 110
Ser Ser Ala Asn Ala Val Gln Ser Ile Cys Asn Ala Leu Gly Val Pro
115 120 125
His Ile Gln Thr Arg Trp Lys His Gln Val Ser Asp Asn Lys Asp Ser
130 135 140
Phe Tyr Val Ser Leu Tyr Pro Asp Phe Ser Ser Leu Ser Arg Ala Ile
145 150 155 160
Leu Asp Leu Val Gln Phe Phe Lys Trp Lys Thr Val Thr Val Val Tyr
165 170 175
Asp Asp Ser Thr Gly Leu Ile Arg Leu Gln Glu Leu Ile Lys Ala Pro
180 185 190
Ser Arg Tyr Asn Leu Arg Leu Lys Ile Arg Gln Leu Pro Ala Asp Thr
195 200 205
Lys Asp Ala Lys Pro Leu Leu Lys Glu Met Lys Arg Gly Lys Glu Phe
210 215 220
His Val Ile Phe Asp Cys Ser His Glu Met Ala Ala Gly Ile Leu Lys
225 230 235 240
Gln Ala Leu Ala Met Gly Met Met Thr Glu Tyr Tyr His Tyr Ile Phe
245 250 255
Thr Thr Leu Asp Leu Phe Ala Leu Asp Val Glu Pro Tyr Arg Tyr Ser
260 265 270
Gly Val Asn Met Thr Gly Phe Arg Ile Leu Asn Thr Glu Asn Thr Gln
275 280 285
Val Ser Ser Ile Ile Glu Lys Trp Ser Met Glu Arg Leu Gln Ala Pro
290 295 300
Pro Lys Pro Asp Ser Gly Leu Leu Asp Gly Phe Met Thr Thr Asp Ala
305 310 315 320
Ala Leu Met Tyr Asp Ala Val His Val Val Ser Val Ala Val Gln Gln
325 330 335
Phe Pro Gln Met Thr Val Ser Ser Leu Gln Cys Asn Arg His Lys Pro
340 345 350
Trp Arg Phe Gly Thr Arg Phe Met Ser Leu Ile Lys Glu Ala His Trp
355 360 365
Glu Gly Leu Thr Gly Arg Ile Thr Phe Asn Lys Thr Asn Gly Leu Arg
370 375 380
Thr Asp Phe Asp Leu Asp Val Ile Ser Leu Lys Glu Glu Gly Leu Glu
385 390 395 400
Lys Ile Gly Thr Trp Asp Pro Ala Ser Gly Leu Asn Met Thr Glu Ser
405 410 415
Gln Lys Gly Lys Pro Ala Asn Ile Thr Asp Ser Leu Ser Asn Arg Ser
420 425 430
Leu Ile Val Thr Thr Ile Leu Glu Glu Pro Tyr Val Leu Phe Lys Lys
435 440 445
Ser Asp Lys Pro Leu Tyr Gly Asn Asp Arg Phe Glu Gly Tyr Cys Ile
450 455 460
Asp Leu Leu Arg Glu Leu Ser Thr Ile Leu Gly Phe Thr Tyr Glu Ile
465 470 475 480
Arg Leu Val Glu Asp Gly Lys Tyr Gly Ala Gln Asp Asp Val Asn Gly
485 490 495
Gln Trp Asn Gly Met Val Arg Glu Leu Ile Asp His Lys Ala Asp Leu
500 505 510
Ala Val Ala Pro Leu Ala Ile Thr Tyr Val Arg Glu Lys Val Ile Asp
515 520 525
Phe Ser Lys Pro Phe Met Thr Leu Gly Ile Ser Ile Leu Tyr Arg Lys
530 535 540
Pro Asn Gly Thr Asn Pro Gly Val Phe Ser Phe Leu Asn Pro Leu Ser
545 550 555 560
Pro Asp Ile Trp Met Tyr Val Leu Leu Ala Cys Leu Gly Val Ser Cys
565 570 575
Val Leu Phe Val Ile Ala Arg Phe Ser Pro Tyr Glu Trp Tyr Asn Pro
580 585 590
His Pro Cys Asn Pro Asp Ser Asp Val Val Glu Asn Asn Phe Thr Leu
595 600 605
Leu Asn Ser Phe Trp Phe Gly Val Gly Ala Leu Met Arg Gln Gly Ser
610 615 620
Glu Leu Met Pro Lys Ala Leu Ser Thr Arg Ile Val Gly Gly Ile Trp
625 630 635 640
Trp Phe Phe Thr Leu Ile Ile Ile Ser Ser Tyr Thr Ala Asn Leu Ala
645 650 655
Ala Phe Leu Thr Val Glu Arg Met Glu Ser Pro Ile Asp Ser Ala Asp
660 665 670
Asp Leu Ala Lys Gln Thr Lys Ile Glu Tyr Gly Ala Val Glu Asp Gly
675 680 685
Ala Thr Met Thr Phe Phe Lys Lys Ser Lys Ile Ser Thr Tyr Asp Lys
690 695 700
Met Trp Ala Phe Met Ser Ser Arg Arg Gln Ser Val Leu Val Lys Ser
705 710 715 720
Asn Glu Glu Gly Ile Gln Arg Val Leu Thr Ser Asp Tyr Ala Phe Leu
725 730 735
Met Glu Ser Thr Thr Ile Glu Phe Val Thr Gln Arg Asn Cys Asn Leu
740 745 750
Thr Gln Ile Gly Gly Leu Ile Asp Ser Lys Gly Tyr Gly Val Gly Thr
755 760 765
Pro Met Gly Ser Pro Tyr Arg Asp Lys Ile Thr Ile Ala Ile Leu Gln
770 775 780
Leu Gln Glu Glu Gly Lys Leu His Met Met Lys Glu Lys Trp Trp Arg
785 790 795 800
Gly Asn Gly Cys Pro Glu Glu Glu Ser Lys Glu Ala Ser Ala Leu Gly
805 810 815
Val Gln Asn Ile Gly Gly Ile Phe Ile Val Leu Ala Ala Gly Leu Val
820 825 830
Leu Ser Val Phe Val Ala Val Gly Glu Phe Leu Tyr Lys Ser Lys Lys
835 840 845
Asn Ala Gln Leu Glu Lys Arg Ser Phe Cys Ser Ala Met Val Glu Glu
850 855 860
Leu Arg Met Ser Leu Lys Cys Gln Arg Arg Leu Lys His Lys Pro Gln
865 870 875 880
Ala Pro Val Ile Val Lys Thr Glu Glu Val Ile Asn Met His Thr Phe
885 890 895
Asn Asp Arg Arg Leu Pro Gly Lys Glu Thr Met Ala
900 905
<210> 115
<211> 4592
<212> DNA
<213> Homo sapiens
<400> 115
gctcgcgcgg ccggacattg tgggtgtgcg tgctggattt ctcccggatg ctctccgact 60
aacatggatg tcccaccatt ccttgcagtg gaaggttgtt ccttggcgca gtgagtgaag 120
aacatgcagc gattgctaat gggtttggga agcggagact ccttcctctc tctatgacca 180
tgccgtgatc gtgtctgcgg tcaccactcg acgcatcctc atttctaccc gaacccagga 240
gccgaacgct agatcgggga agtgggtgcc gtgcgtgtgg gcacagaaac accatgaaga 300
ttattttccc gattctaagt aatccagtct tcaggcgcac cgttaaactc ctgctctgtt 360
tactgtggat tggatattct caaggaacca cacatgtatt aagatttggt ggtatttttg 420
aatatgtgga atctggccca atgggagctg aggaacttgc attcagattt gctgtgaaca 480
caattaacag aaacagaaca ttgctaccca atactaccct tacctatgat acccagaaga 540
taaaccttta tgatagtttt gaagcatcca agaaagcctg tgatcagctg tctcttgggg 600
tggctgccat cttcgggcct tcacacagct catcagcaaa cgcagtgcag tccatctgca 660
atgctctggg agttccccac atacagaccc gctggaagca ccaggtgtca gacaacaaag 720
attccttcta tgtcagtctc tacccagact tctcttcact cagccgtgcc attttagacc 780
tggtgcagtt tttcaagtgg aaaaccgtca cggttgtgta tgatgacagc actggtctca 840
ttcgtttgca agagctcatc aaagctccat caaggtataa tcttcgactc aaaattcgtc 900
agttacctgc tgatacaaag gatgcaaaac ccttactaaa agaaatgaaa agaggcaagg 960
agtttcatgt aatctttgat tgtagccatg aaatggcagc aggcatttta aaacaggcat 1020
tagctatggg aatgatgaca gaatactatc attatatctt taccactctg gacctctttg 1080
ctcttgatgt tgagccctac cgatacagtg gtgttaacat gacagggttc agaatattaa 1140
atacagaaaa tacccaagtc tcctccatca ttgaaaagtg gtcgatggaa cgattgcagg 1200
cacctccgaa acccgattca ggtttgctgg atggatttat gacgactgat gctgctctaa 1260
tgtatgatgc tgtgcatgtg gtgtctgtgg ccgttcaaca gtttccccag atgacagtca 1320
gttccttgca gtgtaatcga cataaaccct ggcgcttcgg gacccgcttt atgagtctaa 1380
ttaaagaggc acattgggaa ggcctcacag gcagaataac tttcaacaaa accaatggct 1440
tgagaacaga ttttgatttg gatgtgatca gtctgaagga agaaggtcta gaaaagattg 1500
gaacgtggga tccagccagt ggcctgaata tgacagaaag tcaaaaggga aagccagcga 1560
acatcacaga ttccttatcc aatcgttctt tgattgttac caccattttg gaagagcctt 1620
atgtcctttt taagaagtct gacaaacctc tctatggtaa tgatcgattt gaaggctatt 1680
gcattgatct cctcagagag ttatctacaa tccttggctt tacatatgaa attagacttg 1740
tggaagatgg gaaatatgga gcccaggatg atgccaatgg acaatggaat ggaatggttc 1800
gtgaactaat tgatcataaa gctgaccttg cagttgctcc actggctatt acctatgttc 1860
gagagaaggt catcgacttt tccaagccct ttatgacact tggaataagt attttgtacc 1920
gcaagcccaa tggtacaaac ccaggcgtct tctccttcct gaatcctctc tcccctgata 1980
tctggatgta tattctgctg gcttacttgg gtgtcagttg tgtgctcttt gtcatagcca 2040
ggtttagtcc ttatgagtgg tataatccac acccttgcaa ccctgactca gacgtggtgg 2100
aaaacaattt taccttgcta aatagtttct ggtttggagt tggagctctc atgcagcaag 2160
gttctgagct catgcccaaa gcactgtcca ccaggatagt gggaggcatt tggtggtttt 2220
tcacacttat catcatttct tcgtatactg ctaacttagc cgcctttctg acagtggaac 2280
gcatggaatc ccctattgac tctgctgatg atttagctaa acaaaccaag atagaatatg 2340
gagcagtaga ggatggtgca accatgactt ttttcaagaa atcaaaaatc tccacgtatg 2400
acaaaatgtg ggcctttatg agtagcagaa ggcagtcagt gctggtcaaa agtaatgaag 2460
aaggaatcca gcgagtcctc acctctgatt atgctttcct aatggagtca acaaccatcg 2520
agtttgttac ccagcggaac tgtaacctga cacagattgg cggccttata gactctaaag 2580
gttatggcgt tggcactccc atgggttctc catatcgaga caaaattacc atagcaattc 2640
ttcagctgca agaggaaggc aaactgcata tgatgaagga gaaatggtgg aggggcaatg 2700
gttgcccaga agaggagagc aaagaggcca gtgccctggg ggttcagaat attggtggca 2760
tcttcattgt tctggcagcc ggcttggtgc tttcagtttt tgtggcagtg ggagaatttt 2820
tatacaaatc caaaaaaaac gctcaattgg aaaagaggtc cttctgtagt gccatggtag 2880
aagaattgag gatgtccctg aagtgccagc gtcggttaaa acataagcca caggccccag 2940
ttattgtgaa aacagaagaa gttatcaaca tgcacacatt taacgacaga aggttgccag 3000
gtaaagaaac catggcataa agctgggagg ccaaacaccc aagcacaaac tgtcgtcttt 3060
ttccaaacaa tttagccaga atgtttcctg tggaaatatg caacctgtgc aaaataaaat 3120
gagttacctc atgccgctgt gtctatgaac tagagactct gtgatctaag cagttgcaat 3180
gatcagactt gatttacaag catcatggat caaccaagtt acacggggtt acactgttaa 3240
tcatgggttc ctcccttctt ctgagtgaat gttaacatgc gcattttgtg gctgatttca 3300
aatgcagtcc agtgagaaat tacaggttcc ttttgaagct caactgttgc caggagatgg 3360
aatatcaatg cccaacaggg caaccaataa aagtgtcact aagaatataa atatttggaa 3420
tcagcaaaaa ctgtagtgtt acaggaaaca gtacagtctt ctgaacaccc agatcataga 3480
ggtgatgatg ttactagccc ccaactactc agtataatta ttgtctgaat gcaaagtatg 3540
tgtttatagg atgtgaaaaa atgtaatgca aaacaaattt gaatcccatg gcagttggaa 3600
tataaagcag atgttcatca cttattttcc ttttttcttt tcttattttt ttttttgaca 3660
gtctgtgtca ctgattgaga tagaaatgcc aattatcaag gaaataatgt tttcttaagt 3720
tccctaaggc agaagattta acatgcaatt ctaccagatc ccttcctatt cccccaacac 3780
cttttctcta acccccatat cccaaataat aataataata ataataataa taataataat 3840
aataataaaa gcagttggtt cagtgattct gaattaaaag gataatgttt tgcaatgttc 3900
aagttgtaaa aactggccga gtattggctg tgtggaagac taaagctttc attctaacat 3960
tcagacatag caatccaaac ccttgttcct gctgtaaatg aacttgatgg agcatgggca 4020
gatttcagtg atacgagaaa ggggactggt catctataga aaaatctgtg agagaacttg 4080
gaagtggact gcgtttatca atacagtcac aatgttaaat gaacaaaatt cttgaacagt 4140
tttttttcaa aaaatgttca ggtttatttg tggaaatgca agatttctat gaaaatagtt 4200
tttgtatgga aatttttgta atacttttta tcaacaaaac aagaacatgt gttcctgtca 4260
ggggtgtgat gtcaagcatg aatggtagtg cgtgtgcacc accaacgttt ggtgaaaact 4320
atttttatca agaaaaaagg aatcatagaa gagaaatatt ttcaagttag ataatataaa 4380
agctaggtgc actaccacca ctgcttacca tgccacaccc ctggtttcca cgaggctgac 4440
aacatactgt aatgaacaat tgtgtgtaaa atggtaaaag acacagacct cttgacaaca 4500
ttgtgataac agttgagtgc acacagtttg ctgtttgaat ccaatgcaca aaattaaaaa 4560
aaatcattaa aactatgttc attttacttt ca 4592
<210> 116
<211> 2727
<212> DNA
<213> Homo sapiens
<400> 116
atgaagatta ttttcccgat tctaagtaat ccagtcttca ggcgcaccgt taaactcctg 60
ctctgtttac tgtggattgg atattctcaa ggaaccacac atgtattaag atttggtggt 120
atttttgaat atgtggaatc tggcccaatg ggagctgagg aacttgcatt cagatttgct 180
gtgaacacaa ttaacagaaa cagaacattg ctacccaata ctacccttac ctatgatacc 240
cagaagataa acctttatga tagttttgaa gcatccaaga aagcctgtga tcagctgtct 300
cttggggtgg ctgccatctt cgggccttca cacagctcat cagcaaacgc agtgcagtcc 360
atctgcaatg ctctgggagt tccccacata cagacccgct ggaagcacca ggtgtcagac 420
aacaaagatt ccttctatgt cagtctctac ccagacttct cttcactcag ccgtgccatt 480
ttagacctgg tgcagttttt caagtggaaa accgtcacgg ttgtgtatga tgacagcact 540
ggtctcattc gtttgcaaga gctcatcaaa gctccatcaa ggtataatct tcgactcaaa 600
attcgtcagt tacctgctga tacaaaggat gcaaaaccct tactaaaaga aatgaaaaga 660
ggcaaggagt ttcatgtaat ctttgattgt agccatgaaa tggcagcagg cattttaaaa 720
caggcattag ctatgggaat gatgacagaa tactatcatt atatctttac cactctggac 780
ctctttgctc ttgatgttga gccctaccga tacagtggtg ttaacatgac agggttcaga 840
atattaaata cagaaaatac ccaagtctcc tccatcattg aaaagtggtc gatggaacga 900
ttgcaggcac ctccgaaacc cgattcaggt ttgctggatg gatttatgac gactgatgct 960
gctctaatgt atgatgctgt gcatgtggtg tctgtggccg ttcaacagtt tccccagatg 1020
acagtcagtt ccttgcagtg taatcgacat aaaccctggc gcttcgggac ccgctttatg 1080
agtctaatta aagaggcaca ttgggaaggc ctcacaggca gaataacttt caacaaaacc 1140
aatggcttga gaacagattt tgatttggat gtgatcagtc tgaaggaaga aggtctagaa 1200
aagattggaa cgtgggatcc agccagtggc ctgaatatga cagaaagtca aaagggaaag 1260
ccagcgaaca tcacagattc cttatccaat cgttctttga ttgttaccac cattttggaa 1320
gagccttatg tcctttttaa gaagtctgac aaacctctct atggtaatga tcgatttgaa 1380
ggctattgca ttgatctcct cagagagtta tctacaatcc ttggctttac atatgaaatt 1440
agacttgtgg aagatgggaa atatggagcc caggatgatg ccaatggaca atggaatgga 1500
atggttcgtg aactaattga tcataaagct gaccttgcag ttgctccact ggctattacc 1560
tatgttcgag agaaggtcat cgacttttcc aagcccttta tgacacttgg aataagtatt 1620
ttgtaccgca agcccaatgg tacaaaccca ggcgtcttct ccttcctgaa tcctctctcc 1680
cctgatatct ggatgtatat tctgctggct tacttgggtg tcagttgtgt gctctttgtc 1740
atagccaggt ttagtcctta tgagtggtat aatccacacc cttgcaaccc tgactcagac 1800
gtggtggaaa acaattttac cttgctaaat agtttctggt ttggagttgg agctctcatg 1860
cagcaaggtt ctgagctcat gcccaaagca ctgtccacca ggatagtggg aggcatttgg 1920
tggtttttca cacttatcat catttcttcg tatactgcta acttagccgc ctttctgaca 1980
gtggaacgca tggaatcccc tattgactct gctgatgatt tagctaaaca aaccaagata 2040
gaatatggag cagtagagga tggtgcaacc atgacttttt tcaagaaatc aaaaatctcc 2100
acgtatgaca aaatgtgggc ctttatgagt agcagaaggc agtcagtgct ggtcaaaagt 2160
aatgaagaag gaatccagcg agtcctcacc tctgattatg ctttcctaat ggagtcaaca 2220
accatcgagt ttgttaccca gcggaactgt aacctgacac agattggcgg ccttatagac 2280
tctaaaggtt atggcgttgg cactcccatg ggttctccat atcgagacaa aattaccata 2340
gcaattcttc agctgcaaga ggaaggcaaa ctgcatatga tgaaggagaa atggtggagg 2400
ggcaatggtt gcccagaaga ggagagcaaa gaggccagtg ccctgggggt tcagaatatt 2460
ggtggcatct tcattgttct ggcagccggc ttggtgcttt cagtttttgt ggcagtggga 2520
gaatttttat acaaatccaa aaaaaacgct caattggaaa agaggtcctt ctgtagtgcc 2580
atggtagaag aattgaggat gtccctgaag tgccagcgtc ggttaaaaca taagccacag 2640
gccccagtta ttgtgaaaac agaagaagtt atcaacatgc acacatttaa cgacagaagg 2700
ttgccaggta aagaaaccat ggcataa 2727
<210> 117
<211> 2610
<212> DNA
<213> Homo sapiens
<400> 117
atgaagatta ttttcccgat tctaagtaat ccagtcttca ggcgcaccgt taaactcctg 60
ctctgtttac tgtggattgg atattctcaa ggaaccacac atgtattaag atttggtggt 120
atttttgaat atgtggaatc tggcccaatg ggagctgagg aacttgcatt cagatttgct 180
gtgaacacaa ttaacagaaa cagaacattg ctacccaata ctacccttac ctatgatacc 240
cagaagataa acctttatga tagttttgaa gcatccaaga aagcctgtga tcagctgtct 300
cttggggtgg ctgccatctt cgggccttca cacagctcat cagcaaacgc agtgcagtcc 360
atctgcaatg ctctgggagt tccccacata cagacccgct ggaagcacca ggtgtcagac 420
aacaaagatt ccttctatgt cagtctctac ccagacttct cttcactcag ccgtgccatt 480
ttagacctgg tgcagttttt caagtggaaa accgtcacgg ttgtgtatga tgacagcact 540
ggtctcattc gtttgcaaga gctcatcaaa gctccatcaa ggtataatct tcgactcaaa 600
attcgtcagt tacctgctga tacaaaggat gcaaaaccct tactaaaaga aatgaaaaga 660
ggcaaggagt ttcatgtaat ctttgattgt agccatgaaa tggcagcagg cattttaaaa 720
caggcattag ctatgggaat gatgacagaa tactatcatt atatctttac cactctggac 780
ctctttgctc ttgatgttga gccctaccga tacagtggtg ttaacatgac agggttcaga 840
atattaaata cagaaaatac ccaagtctcc tccatcattg aaaagtggtc gatggaacga 900
ttgcaggcac ctccgaaacc cgattcaggt ttgctggatg gatttatgac gactgatgct 960
gctctaatgt atgatgctgt gcatgtggtg tctgtggccg ttcaacagtt tccccagatg 1020
acagtcagtt ccttgcagtg taatcgacat aaaccctggc gcttcgggac ccgctttatg 1080
agtctaatta aagaggcaca ttgggaaggc ctcacaggca gaataacttt caacaaaacc 1140
aatggcttga gaacagattt tgatttggat gtgatcagtc tgaaggaaga aggtctagaa 1200
aagattggaa cgtgggatcc agccagtggc ctgaatatga cagaaagtca aaagggaaag 1260
ccagcgaaca tcacagattc cttatccaat cgttctttga ttgttaccac cattttggaa 1320
gagccttatg tcctttttaa gaagtctgac aaacctctct atggtaatga tcgatttgaa 1380
ggctattgca ttgatctcct cagagagtta tctacaatcc ttggctttac atatgaaatt 1440
agacttgtgg aagatgggaa atatggagcc caggatgatg ccaatggaca atggaatgga 1500
atggttcgtg aactaattga tcataaagct gaccttgcag ttgctccact ggctattacc 1560
tatgttcgag agaaggtcat cgacttttcc aagcccttta tgacacttgg aataagtatt 1620
ttgtaccgca agcccaatgg tacaaaccca ggcgtcttct ccttcctgaa tcctctctcc 1680
cctgatatct ggatgtatat tctgctggct tacttgggtg tcagttgtgt gctctttgtc 1740
atagccaggt ttagtcctta tgagtggtat aatccacacc cttgcaaccc tgactcagac 1800
gtggtggaaa acaattttac cttgctaaat agtttctggt ttggagttgg agctctcatg 1860
cagcaaggtt ctgagctcat gcccaaagca ctgtccacca ggatagtggg aggcatttgg 1920
tggtttttca cacttatcat catttcttcg tatactgcta acttagccgc ctttctgaca 1980
gtggaacgca tggaatcccc tattgactct gctgatgatt tagctaaaca aaccaagata 2040
gaatatggag cagtagagga tggtgcaacc atgacttttt tcaagaaatc aaaaatctcc 2100
acgtatgaca aaatgtgggc ctttatgagt agcagaaggc agtcagtgct ggtcaaaagt 2160
aatgaagaag gaatccagcg agtcctcacc tctgattatg ctttcctaat ggagtcaaca 2220
accatcgagt ttgttaccca gcggaactgt aacctgacac agattggcgg ccttatagac 2280
tctaaaggtt atggcgttgg cactcccatg ggttctccat atcgagacaa aattaccata 2340
gcaattcttc agctgcaaga ggaaggcaaa ctgcatatga tgaaggagaa atggtggagg 2400
ggcaatggtt gcccagaaga ggagagcaaa gaggccagtg ccctgggggt tcagaatatt 2460
ggtggcatct tcattgttct ggcagccggc ttggtgcttt cagtttttgt ggcagtggga 2520
gaatttttat acaaatccaa aaaaaacgct caattggaaa aggaatcttc tatttggtta 2580
gtgccaccat accatccaga cactgtttag 2610
<210> 118
<211> 2679
<212> DNA
<213> Homo sapiens
<400> 118
atgaagatta ttttcccgat tctaagtaat ccagtcttca ggcgcaccgt taaactcctg 60
ctctgtttac tgtggattgg atattctcaa ggaaccacac atgtattaag atttggtggt 120
atttttgaat atgtggaatc tggcccaatg ggagctgagg aacttgcatt cagatttgct 180
gtgaacacaa ttaacagaaa cagaacattg ctacccaata ctacccttac ctatgatacc 240
cagaagataa acctttatga tagttttgaa gcatccaaga aagcctgtga tcagctgtct 300
cttggggtgg ctgccatctt cgggccttca cacagctcat cagcaaacgc agtgcagtcc 360
atctgcaatg ctctgggagt tccccacata cagacccgct ggaagcacca ggtgtcagac 420
aacaaagatt ccttctatgt cagtctctac ccagacttct cttcactcag ccgtgccatt 480
ttagacctgg tgcagttttt caagtggaaa accgtcacgg ttgtgtatga tgacagcact 540
ggtctcattc gtttgcaaga gctcatcaaa gctccatcaa ggtataatct tcgactcaaa 600
attcgtcagt tacctgctga tacaaaggat gcaaaaccct tactaaaaga aatgaaaaga 660
ggcaaggagt ttcatgtaat ctttgattgt agccatgaaa tggcagcagg cattttaaaa 720
caggcattag ctatgggaat gatgacagaa tactatcatt atatctttac cactctggac 780
ctctttgctc ttgatgttga gccctaccga tacagtggtg ttaacatgac agggttcaga 840
atattaaata cagaaaatac ccaagtctcc tccatcattg aaaagtggtc gatggaacga 900
ttgcaggcac ctccgaaacc cgattcaggt ttgctggatg gatttatgac gactgatgct 960
gctctaatgt atgatgctgt gcatgtggtg tctgtggccg ttcaacagtt tccccagatg 1020
acagtcagtt ccttgcagtg taatcgacat aaaccctggc gcttcgggac ccgctttatg 1080
agtctaatta aagaggcaca ttgggaaggc ctcacaggca gaataacttt caacaaaacc 1140
aatggcttga gaacagattt tgatttggat gtgatcagtc tgaaggaaga aggtctagaa 1200
aagattggaa cgtgggatcc agccagtggc ctgaatatga cagaaagtca aaagggaaag 1260
ccagcgaaca tcacagattc cttatccaat cgttctttga ttgttaccac cattttggaa 1320
gagccttatg tcctttttaa gaagtctgac aaacctctct atggtaatga tcgatttgaa 1380
ggctattgca ttgatctcct cagagagtta tctacaatcc ttggctttac atatgaaatt 1440
agacttgtgg aagatgggaa atatggagcc caggatgatg ccaatggaca atggaatgga 1500
atggttcgtg aactaattga tcataaagct gaccttgcag ttgctccact ggctattacc 1560
tatgttcgag agaaggtcat cgacttttcc aagcccttta tgacacttgg aataagtatt 1620
ttgtaccgca agcccaatgg tacaaaccca ggcgtcttct ccttcctgaa tcctctctcc 1680
cctgatatct ggatgtatat tctgctggct tacttgggtg tcagttgtgt gctctttgtc 1740
atagccaggt ttagtcctta tgagtggtat aatccacacc cttgcaaccc tgactcagac 1800
gtggtggaaa acaattttac cttgctaaat agtttctggt ttggagttgg agctctcatg 1860
cagcaaggtt ctgagctcat gcccaaagca ctgtccacca ggatagtggg aggcatttgg 1920
tggtttttca cacttatcat catttcttcg tatactgcta acttagccgc ctttctgaca 1980
gtggaacgca tggaatcccc tattgactct gctgatgatt tagctaaaca aaccaagata 2040
gaatatggag cagtagagga tggtgcaacc atgacttttt tcaagaaatc aaaaatctcc 2100
acgtatgaca aaatgtgggc ctttatgagt agcagaaggc agtcagtgct ggtcaaaagt 2160
aatgaagaag gaatccagcg agtcctcacc tctgattatg ctttcctaat ggagtcaaca 2220
accatcgagt ttgttaccca gcggaactgt aacctgacac agattggcgg ccttatagac 2280
tctaaaggtt atggcgttgg cactcccatg ggttctccat atcgagacaa aattaccata 2340
gcaattcttc agctgcaaga ggaaggcaaa ctgcatatga tgaaggagaa atggtggagg 2400
ggcaatggtt gcccagaaga ggagagcaaa gaggccagtg ccctgggggt tcagaatatt 2460
ggtggcatct tcattgttct ggcagccggc ttggtgcttt cagtttttgt ggcagtggga 2520
gaatttttat acaaatccaa aaaaaacgct caattggaaa agagagccaa gactaagtta 2580
cctcaagact atgtattcct ccctattttg gagtcagttt ccatttctac agtgttgtca 2640
tcatcaccat cttcatcatc attatcatca tgttcttaa 2679
<210> 119
<211> 10568
<212> DNA
<213> Homo sapiens
<400> 119
acttgcgctc tcgctggcgg ctgcgtggcc gaggctggga gcccgggact tcccgctgaa 60
ccgcctcctg ccgcagctct gagaggacta ccccagtccc taccctccct cttcacccta 120
gccgcaggct cgcgcggctg gacattgtgc ttgctggatt tttcccggat gctcccagac 180
taacatggat gtcccaccat cccttgcagt ggaagcttgc tccttggcgc agtgagtgaa 240
gaacatgcag agactgctaa tgggtttggg aagcggagac tccttcctct ttctgtgacc 300
atgccgtgat tgtgtttgcg gccactattc cacgcatcct tcttctcgtc caagcccgga 360
gcctaacgct agatcgggga agtgggtgcc gcgcgcgcag gcacggaaac atcatgaaga 420
ttatttcccc agttttaagt aatctagtct tcagtcgctc cattaaagtc ctgctctgct 480
tgttgtggat cggatattcg caaggaacca cacatgtgtt aagattcggt ggtatatttg 540
aatatgtgga atctggccct atgggagctg aagaacttgc attcagattt gctgtgaata 600
caatcaacag gaacaggact ctgctaccca ataccacgtt aacatatgat acacagaaga 660
tcaatctcta tgacagtttt gaagcatcta agaaagcttg tgatcagctg tctcttgggg 720
tggctgccat cttcggtcct tcacacagtt catcagcaaa tgctgttcag tccatctgca 780
atgctctggg ggttcctcac atacagaccc gctggaagca ccaggtgtca gacaataagg 840
attccttcta tgtcagtctc tacccagact tctcttccct cagccgtgcc atcttggatt 900
tggtgcagtt ttttaagtgg aaaactgtca cagttgtgta tgacgacagc actggtctca 960
ttcgcttgca agagctcatc aaagctccat caaggtacaa tcttcgactt aaaattcgtc 1020
agctgccagc tgatacaaaa gatgcaaagc ctttgctgaa agagatgaag aggggcaagg 1080
agttccacgt gatcttcgac tgcagccatg aaatggcagc aggcatttta aagcaggcat 1140
tagctatggg aatgatgaca gaatactacc actatatatt tacgactctg gacctcttcg 1200
ctcttgatgt ggagccctac agatacagtg gcgtaaatat gacagggttc agaatactaa 1260
atacagagaa tacccaagtc tcctccatca tcgagaagtg gtcgatggaa cggttacagg 1320
cacctccaaa acctgactca ggtttgctgg atggatttat gacgactgat gctgctctga 1380
tgtatgatgc agtgcacgtt gtgtctgtag ctgtccaaca gtttccccag atgacagtca 1440
gctccttgca atgcaatcga cacaaaccct ggcgctttgg gactcgcttc atgagcctaa 1500
ttaaagaggc tcattgggaa ggtcttacag gcagaattac atttaacaaa accaatggat 1560
tgcgaacaga ttttgatttg gatgtgatca gtctcaagga agaaggtctg gagaagattg 1620
ggacttggga tccatccagt ggcctgaata tgacagaaag tcagaaaggg aagccagcaa 1680
atattacaga ttcattgtct aatcgttctt tgattgttac caccattttg gaagaaccat 1740
atgtcctgtt taagaagtct gacaaacctc tctatgggaa tgatcgattt gaaggctact 1800
gtattgatct tctacgagag ttatctacaa tccttggctt tacatatgaa attaggcttg 1860
tggaggatgg gaaatatgga gcccaggatg atgtgaatgg acaatggaat ggaatggttc 1920
gtgagctaat tgatcataaa gctgaccttg cagttgctcc actggctatt acctatgttc 1980
gtgagaaggt catcgacttt tcaaagccgt ttatgactct tggaataagt attttgtacc 2040
gcaagcccaa tggtacaaac ccaggcgtct tctccttcct gaatcctctc tcccctgata 2100
tctggatgta tattctgctg gcttacttgg gtgtcagttg tgtgctcttt gtcatagcca 2160
ggtttagtcc ctatgagtgg tataatccac acccttgcaa ccctgactca gacgtggtgg 2220
aaaacaattt taccttgcta aatagtttct ggtttggagt tggagctctc atgcagcaag 2280
gttctgagct catgcccaaa gcactctcca ccaggatagt gggaggcatt tggtggtttt 2340
tcacacttat catcatttct tcgtataccg ctaacctagc cgcctttctg accgtggaac 2400
gcatggagtc gcctattgac tctgctgacg atttagctaa gcaaaccaag atagaatatg 2460
gagcagtaga ggacggcgca accatgacgt ttttcaagaa atcaaaaatc tcaacgtatg 2520
ataaaatgtg ggcatttatg agcagcagga gacagtctgt gcttgtcaaa agcaatgagg 2580
aagggattca acgtgtcctc acctccgatt atgctttctt aatggagtca acgaccatcg 2640
agtttgttac ccagcggaac tgtaacctca cgcagattgg tggccttata gactccaaag 2700
gctatggtgt tggcactccc atgggttctc catatcgaga caaaatcacc atagccattc 2760
ttcagctgca ggaggaaggc aagctgcaca tgatgaagga gaagtggtgg cgaggcaatg 2820
gctgcccaga ggaggagagc aaagaggcca gtgctctagg ggtgcagaat attggtggta 2880
tcttcattgt cctggcagcc ggcttggtgc tctcagtttt tgtggcagtg ggagagtttt 2940
tatacaaatc caaaaaaaac gctcaattgg aaaagaggtc cttctgtagc gccatggtgg 3000
aagaactgag aatgtctctg aagtgccagc gtcggctcaa acataagcca caggccccag 3060
ttattgtgaa aacagaagaa gttatcaaca tgcacacatt taacgacaga aggttgccag 3120
gtaaagaaac catggcatga agctgggagg ccaatcaccc aagcacaaac tgtcgtcttt 3180
tttttttttt tcaaacaatt tagcgagaat gtttcctgtg gaaatatgca acctgtgcaa 3240
aataaaatga gttacctcat gccgctgtgt ctatgaacta gagactcttg tgatctaagc 3300
agtttcagtg atcagacttg atttacaagc accatggatc gacaaagtta cacggggtta 3360
cactgtttat catgggttcc tcccttcctt tgagtgaatg ttacatgaaa atgttgtggc 3420
tggtttcaaa tgcagtccag agagaaactg ctggttcctt ctgaagctca actgttgtca 3480
ggagatggaa tgttggggcc caaaaggata accaataaaa atgccataat ttataaaagc 3540
aaaacaaaaa gcgtgtgaaa tctgcaaaaa ttgtagtgtc acaagaaaca gtatagtccc 3600
atggtcacca acaaaatgag gtgataatgt tactagcccc caatactcag taaaatcatc 3660
atctgaatag ataatgtgtt catagaatgt ggaaaaaatg taatgcaaaa catatcagta 3720
ttcaatcaaa gtggaacaga aagcagacca ccatcagtta ttttcctttc tcaatagtct 3780
gtgtcatgga ttgtgatata gatggcaatt atctatctaa ttgttttctt aaaataccca 3840
tggcaaatat tttaaaatgc aacttgctcc caggaacccc taccctaacc tacactagaa 3900
ataaaaaagc caccactggt ataaagattc tgatgtaaaa gatatgtttt tcaatccttg 3960
tcatgaattg taaaacaggg ctcagtatta ctggttatat ggaagactga agctttcact 4020
ctgacattct gatatgtcag ctgaaactct ccttcctcct ggaaaggacc ttgatggagc 4080
ctgggcagat tccattgata agactgggga cttgtcacct atacagaact acgtgacaga 4140
actttgaggt ggactgcatt taacaatagt cacaatgtta aaagaacaaa attcttgagc 4200
agtttttttt ttctgttttg ttttaaaaaa tgttcaggtt tatttgtgga aatgcaagat 4260
ttctatgaaa atagtttttg tatggaaatt tttgtaatac tttttatcaa caaaataaga 4320
acatgtgttc ctgtcagggg tgtgatgtca agcatgaacg gtagtgcgtg tgcaccacca 4380
acgtttggtg aaaactattt ttatcaagaa aaaggaatca tagaagagaa atattttcaa 4440
gttagatact ataaaagcta ggtgcactac caccacggct tgtcacgcca cacccctgag 4500
tcccacaagg cagataacat attgtaatga acagttgtgt gtaaaatgat aaaagacaca 4560
gacctcttga caacattgtg aaaacagttg agtgcacaca gtttgctgtt tgaatccaat 4620
gcacaaaaat tttacaaaac tccattaaaa ttatgtccat tttactttca gctttggctt 4680
tgatttttct cttgcatgtg taaatgaatg taacatggtg gttttgtata gaaaatatac 4740
atcaaggggt cttaggatct caaagttaga atcttcccaa cttacagcaa aaaggaaaag 4800
gccatcctgg aggtgctcct ctctttctct ctcccttcct ctgtctctct ctttgactct 4860
gtctctttgt gtctcagtct ttctctatat cagtttttct ctgtccctct ctacctctgg 4920
ctctatcact ctctgtctca tttacacaca tacacacaca cacacacaca cacacacaca 4980
cacgaataaa gacatataca ttggttttag aattagggta gctggaataa aaagaatatg 5040
attgtagaga tggcaacctt tatcttatct catttgtagc tggaaattga ctaagttcac 5100
tgtgctgcat tatgttgtgg aatggtaatt atctactttg gttcaactca tatccaattt 5160
cagaattttc tgtgcattga tacttcaata atcatcagca gaggaacaaa aagggaaaag 5220
tttagaatta ataattaatt ttagatccta acatattaat agaaacaaac tataacagtt 5280
ttacgttttg aaaatcaaat ctgtaagatt caacttattt tcctgattaa ttaattaatt 5340
aattctaagt gtgcaattat aattggaatc tgacaaaaaa aaaaccccac tggaaaagtt 5400
tccataatgt atttcttaaa atagtaaaaa ttgcataatc aaattatctc aaattaatta 5460
agatttatat atgtgagcac tttaaatatt ttatgctatg attattatca gatttcaatg 5520
attattttgt tcaagctaca aatgtagcta tacaaatcac tgctaaagta gcagtactgg 5580
tgtattagtg ccaaccaagt ttaaataagg aaaataatat aagtattcag attattaaga 5640
tgctgttttt aacaaacaaa ttttaaattt tatgaaaata tgaatttgta aaggaaacaa 5700
acttcattat taatattatg gggaaattct gtgaatatat acaatctgac acatgtagaa 5760
tttgcacatt cgatgagaaa ctgtgtcaaa aatgatcaat tgaagcaact catttaataa 5820
aaaagaactc tcactaagca atgctttaat atgttttaaa gatataattt taaacacttg 5880
gaaatcttat atatgtgagt ataaaaacac attaaatatt catatcttaa tttacttaaa 5940
agagttttaa cttcaattta ttactgaatt taataatcat aaacattggg tataaaatta 6000
caactttatt gttaagcagc cagaagagaa tgaattctgg taaaatttga taatgatttt 6060
attgaaaaat tgaattaagt tcttaaatat gtaacaacct tactagacat taactttata 6120
aatgatatta aatgtttata atatattctt ttacattcta attctaatta atacttcata 6180
catgtataag tacttaaatt ttccaaaaca cctggtcata atatatatat taattttaat 6240
tataataaaa atgctaatta ctttcataca taatatcaaa caatgaaaaa actctaggtt 6300
ggaagcatgt aagagttctc agcattttct agaggaaaaa tcaaaagcaa agagaagcta 6360
atacctgttc taggcaacat cagaacctat gaattgagag gatgaatgga ggctcatgga 6420
cttcagctat agaaaccaac aaggatggag acaccagagg ttttataccc acatttacta 6480
ccactaaata atcagtatct ccctgggaac accaaagaaa cacacataat taccactgaa 6540
caaccctaac tctcatggga acacaagaga aatatagtct agtttataaa tgtgtaaaat 6600
gaagaatagt ttattgatgg gaatatttca gatattttgt tgctcactta tatctacata 6660
taagtgcata tgtaataaaa attcatatcc catggcacca ttcttgggtg gtatagatat 6720
gaaacattgc tggtttcaaa atgtttaagg attgtgtcaa cttttgatgt ctgtttactt 6780
tgaaatatat tgtaactgca gaaaagtgca tatacaaggc atatatacac agaattgctg 6840
ctagttgtta tagttttaaa cacagttatt gtgccattta acccaggctc aatgaaagtt 6900
ctctgacttg ctgataacat tttcagggaa gaaattacag tgatctttga tcatgaaatg 6960
tataattaac tcatctgtgt ttgactatgc caatagagtg tgtacagcat tgtagagaaa 7020
cagtcttggg attttgtggc agcttttaaa ggtagaatca tagcaatgaa aactttggtt 7080
atgcctgtcc tacctaaatg tatacaaaat ttgaaccatt ttcctccatt gtgagctgat 7140
acgtgaccag atctgcagac atgtaaaatt gtgaagttat tcccttgtct ttaggtattt 7200
gcctcaatgc aaattcaaat gtaataattt tttttatttc tcttaaaata tgtcagttaa 7260
ttatttgatt ttttctcaat tttggtggtt ttatagaaat gctgaatagg ctcaatgggt 7320
tttagatgtt ttatgtctat tattctactc tagtctataa ttatctggat attcattgac 7380
tcctagctca aaattagttc cttggaaaat ttgaagactt tccactattt cctcttcccc 7440
ctcatctatt tcccattttc tcccccattt ccatctcttt cccccattcc tcatccttta 7500
tatatatata tatatatata tatacatata tatatatata tatgtgtgtg tgtgtgtgtg 7560
tgtgtgtgtg tgtgtgtgtg tgtgtgtact tgctttttta ttatcttgtc taatgttgag 7620
attgctgtgg aaaacctaaa atcatgatca gatggtcaca gttctttgat catcttcttc 7680
ttcttcttct tcttcttctt cttcttcttc ttcttcttct tcttcttctt cttcttcttc 7740
ttcttcttct tcctcttcct cctcctcctc ctcttcctcc tcctcttcct cttcgtcgtc 7800
ttctccttct cctccttctt ttccttctct tcctccacct cttcctttct tcttcttttc 7860
cctcttcttc ctccttctct tgtgcctcct ctttttgttt ttcttacttc tctctttgtc 7920
tctatctcta tgtttctgtt tctttgtctc tgtctctctc tctgtgtctc tgtctgtcag 7980
tctctgtcgg cctgtctgtc tttctctctg tctcagatgt agattgatct gtatcaattg 8040
tactaaccct tactagactc cttaataaag ctactatttt tcaattttaa gaaaagacct 8100
tgaaataata cttcattgtc ttcctttctt catatttcct ttgttctctc taaatctgtt 8160
cttatgactt gtaattactt aactttgaat cttcagtcta tttacttttc tatttctcta 8220
tattgttatt ttaagtctat aatctaaaat acttttttag tttctcctat ctttgacatt 8280
gttttaattt ctgtaagtta ctgtttaatt cctatgacat cttcacttat atcatagtag 8340
ttgatcataa ttgttcttta ttcccatcat taggaaaaga cattgtatat tcctgttctt 8400
ttctattttt ttcacctgag ttgaaggtat tattgagata acagttgagt catttaccct 8460
ccagttactc ctcccagttc ttttcacttt ctctcccaca tgtatctact tcctgtttgt 8520
ctctcattca aaaaggaaag gcctctaaga gataacagca aaatttaaca acataaaata 8580
taataacata aaacacacaa gcaaaaactg tcacatcaaa gttgaacaag ataaaccaac 8640
aaaacaaaaa gagcctcaag aaggcagaag aatcagagag cctcttgttc acttacctca 8700
gttgaattag aattggttct tatcctgtgg gtagtgaccc atttagaggt agaatgaccc 8760
tttcttaaag ttttcctaag acaatcagac aatatagatg tttacattat gatttataaa 8820
tagcaaaatt atagttatga aatagaggca aaaataattt tatggttttg ggtcattaca 8880
acattaagaa ccatatttga gggttctagc cttaggaagg ttgaaaacca ctgaattata 8940
aagggccttg tctccttgtg tcctctaccc ttttgaatct ttcacatttc tcacctcatc 9000
ttctgtgggt gggatttagt ggagacatcc cactgaaagc tgtgttccaa atgcatctgt 9060
ttgttaaatg tcatccgtca actcttactg ttctctttat gatgatgtaa agtagaataa 9120
acatcactgg caaatttttg tttatgttaa gttgtgacag aatgttcatc taacttgagt 9180
aatcctgtag gtgaatatat gtggactact tgatgaataa tctgtccaag tacctccgtt 9240
tggtttttac ttcttgggta tattagattt gcagagaggt gtcttttaac ttcattccta 9300
atgccttgaa atcctgatca ctatatttca gccaccagag tcaagaactt atcactgcaa 9360
attctgccct gtgtgtgagt gaataaagca atttttgtcc catttattct gaaaatttgc 9420
gtttaattat atctcacatt cttcatacta gaatatacat tttcctcttt taaaaatatg 9480
cacacctgac agactgtgtt tgaaatagtg ggaacaccat actctatggt tccagtgtct 9540
gttttgacca tcacattact atcttctctc ccaagggcca agtctaccaa gtggaatatg 9600
cctttaatgc tattaaccag gctggtctta tacttgtagc tgtcagagga aaagactgct 9660
caggttttgt cacatagatg aaagtacatg acaaattact agattccaac acacttgttg 9720
aagataactg aaaccagtgg ttgtataatg atgggagtaa cagctgacag actccaggta 9780
caaatggcac actatgaggt agttaattgg aaagacaaag gtaactatga gtttcctgtg 9840
gacatgctat gtaaaagaat tgctgatatt actcaagtca acacacagaa taatgaaatg 9900
aggctgcttc attgttgttt gatttttttt tactggtata aatgaaaaaa caaggccctc 9960
aagtgtacta gtggaatcct ataggtgtta agccatgaca gcagaagtta aatgaatata 10020
accaaccagc ttccttgaaa acaataagtg aggaagaaaa ttgattggat atttgaataa 10080
acatttagat aatgtaatta catgtctgcc tactattctg tcgattggtt tcaaactttc 10140
agaaatagaa attggaagag ttagagttga aaatactcga ttcaggattc ttacagaagc 10200
agagattggc actcatcttg ttgttctagc agagagactg aacattgtca tcagtttacc 10260
aaatctgtga tgccacttgc ctgtgtgttt gataaaaacc aacatcatag aggctccaca 10320
gcttaaaatg gaacctcttc cactcctgcc actgagctgc ttaggactct gtataaataa 10380
aaacagtcct tttggaaaaa taaatatgta cactgtactt aaaaataaac acatgaaatt 10440
tttatgtgct acgttaaaac ctaactccaa aatttaaaga aacgctacaa ttgtccacat 10500
ccattaaaag actccttgtt attttatgtt ctcttttgta ccactattaa attgattctc 10560
tattgcta 10568
<210> 120
<211> 10655
<212> DNA
<213> Mus musculus
<400> 120
acttgcgctc tcgctggcgg ctgcgtggcc gaggctggga gcccgggact tcccgctgaa 60
ccgcctcctg ccgcagctct gagaggacta ccccagtccc taccctccct cttcacccta 120
gccgcaggct cgcgcggctg gacattgtgc ttgctggatt tttcccggat gctcccagac 180
taacatggat gtcccaccat cccttgcagt ggaagcttgc tccttggcgc agtgagtgaa 240
gaacatgcag agactgctaa tgggtttggg aagcggagac tccttcctct ttctgtgacc 300
atgccgtgat tgtgtttgcg gccactattc cacgcatcct tcttctcgtc caagcccgga 360
gcctaacgct agatcgggga agtgggtgcc gcgcgcgcag gcacggaaac atcatgaaga 420
ttatttcccc agttttaagt aatctagtct tcagtcgctc cattaaagtc ctgctctgct 480
tgttgtggat cggatattcg caaggaacca cacatgtgtt aagattcggt ggtatatttg 540
aatatgtgga atctggccct atgggagctg aagaacttgc attcagattt gctgtgaata 600
caatcaacag gaacaggact ctgctaccca ataccacgtt aacatatgat acacagaaga 660
tcaatctcta tgacagtttt gaagcatcta agaaagcttg tgatcagctg tctcttgggg 720
tggctgccat cttcggtcct tcacacagtt catcagcaaa tgctgttcag tccatctgca 780
atgctctggg ggttcctcac atacagaccc gctggaagca ccaggtgtca gacaataagg 840
attccttcta tgtcagtctc tacccagact tctcttccct cagccgtgcc atcttggatt 900
tggtgcagtt ttttaagtgg aaaactgtca cagttgtgta tgacgacagc actggtctca 960
ttcgcttgca agagctcatc aaagctccat caaggtacaa tcttcgactt aaaattcgtc 1020
agctgccagc tgatacaaaa gatgcaaagc ctttgctgaa agagatgaag aggggcaagg 1080
agttccacgt gatcttcgac tgcagccatg aaatggcagc aggcatttta aagcaggcat 1140
tagctatggg aatgatgaca gaatactacc actatatatt tacgactctg gacctcttcg 1200
ctcttgatgt ggagccctac agatacagtg gcgtaaatat gacagggttc agaatactaa 1260
atacagagaa tacccaagtc tcctccatca tcgagaagtg gtcgatggaa cggttacagg 1320
cacctccaaa acctgactca ggtttgctgg atggatttat gacgactgat gctgctctga 1380
tgtatgatgc agtgcacgtt gtgtctgtag ctgtccaaca gtttccccag atgacagtca 1440
gctccttgca atgcaatcga cacaaaccct ggcgctttgg gactcgcttc atgagcctaa 1500
ttaaagaggc tcattgggaa ggtcttacag gcagaattac atttaacaaa accaatggat 1560
tgcgaacaga ttttgatttg gatgtgatca gtctcaagga agaaggtctg gagaagattg 1620
ggacttggga tccatccagt ggcctgaata tgacagaaag tcagaaaggg aagccagcaa 1680
atattacaga ttcattgtct aatcgttctt tgattgttac caccattttg gaagaaccat 1740
atgtcctgtt taagaagtct gacaaacctc tctatgggaa tgatcgattt gaaggctact 1800
gtattgatct tctacgagag ttatctacaa tccttggctt tacatatgaa attaggcttg 1860
tggaggatgg gaaatatgga gcccaggatg atgtgaatgg acaatggaat ggaatggttc 1920
gtgagctaat tgatcataaa gctgaccttg cagttgctcc actggctatt acctatgttc 1980
gtgagaaggt catcgacttt tcaaagccgt ttatgactct tggaataagt attttgtacc 2040
gcaagcccaa tggtacaaac ccaggcgtct tctccttcct gaatcctctc tcccctgata 2100
tctggatgta tattctgctg gcttacttgg gtgtcagttg tgtgctcttt gtcatagcca 2160
ggtttagtcc ctatgagtgg tataatccac acccttgcaa ccctgactca gacgtggtgg 2220
aaaacaattt taccttgcta aatagtttct ggtttggagt tggagctctc atgcagcaag 2280
gttctgagct catgcccaaa gcactctcca ccaggatagt gggaggcatt tggtggtttt 2340
tcacacttat catcatttct tcgtataccg ctaacctagc cgcctttctg accgtggaac 2400
gcatggagtc gcctattgac tctgctgacg atttagctaa gcaaaccaag atagaatatg 2460
gagcagtaga ggacggcgca accatgacgt ttttcaagaa atcaaaaatc tcaacgtatg 2520
ataaaatgtg ggcatttatg agcagcagga gacagtctgt gcttgtcaaa agcaatgagg 2580
aagggattca acgtgtcctc acctccgatt atgctttctt aatggagtca acgaccatcg 2640
agtttgttac ccagcggaac tgtaacctca cgcagattgg tggccttata gactccaaag 2700
gctatggtgt tggcactccc atgggttctc catatcgaga caaaatcacc atagccattc 2760
ttcagctgca ggaggaaggc aagctgcaca tgatgaagga gaagtggtgg cgaggcaatg 2820
gctgcccaga ggaggagagc aaagaggcca gtgctctagg ggtgcagaat attggtggta 2880
tcttcattgt cctggcagcc ggcttggtgc tctcagtttt tgtggcagtg ggagagtttt 2940
tatacaaatc caaaaaaaac gctcaattgg aaaaggaatc ttctatttgg ttagtgccac 3000
cataccatcc agacactgtt tagtaaactt ttgaaacttt ctaaaagagg tttttaatga 3060
tgaggtcctt ctgtagcgcc atggtggaag aactgagaat gtctctgaag tgccagcgtc 3120
ggctcaaaca taagccacag gccccagtta ttgtgaaaac agaagaagtt atcaacatgc 3180
acacatttaa cgacagaagg ttgccaggta aagaaaccat ggcatgaagc tgggaggcca 3240
atcacccaag cacaaactgt cgtctttttt ttttttttca aacaatttag cgagaatgtt 3300
tcctgtggaa atatgcaacc tgtgcaaaat aaaatgagtt acctcatgcc gctgtgtcta 3360
tgaactagag actcttgtga tctaagcagt ttcagtgatc agacttgatt tacaagcacc 3420
atggatcgac aaagttacac ggggttacac tgtttatcat gggttcctcc cttcctttga 3480
gtgaatgtta catgaaaatg ttgtggctgg tttcaaatgc agtccagaga gaaactgctg 3540
gttccttctg aagctcaact gttgtcagga gatggaatgt tggggcccaa aaggataacc 3600
aataaaaatg ccataattta taaaagcaaa acaaaaagcg tgtgaaatct gcaaaaattg 3660
tagtgtcaca agaaacagta tagtcccatg gtcaccaaca aaatgaggtg ataatgttac 3720
tagcccccaa tactcagtaa aatcatcatc tgaatagata atgtgttcat agaatgtgga 3780
aaaaatgtaa tgcaaaacat atcagtattc aatcaaagtg gaacagaaag cagaccacca 3840
tcagttattt tcctttctca atagtctgtg tcatggattg tgatatagat ggcaattatc 3900
tatctaattg ttttcttaaa atacccatgg caaatatttt aaaatgcaac ttgctcccag 3960
gaacccctac cctaacctac actagaaata aaaaagccac cactggtata aagattctga 4020
tgtaaaagat atgtttttca atccttgtca tgaattgtaa aacagggctc agtattactg 4080
gttatatgga agactgaagc tttcactctg acattctgat atgtcagctg aaactctcct 4140
tcctcctgga aaggaccttg atggagcctg ggcagattcc attgataaga ctggggactt 4200
gtcacctata cagaactacg tgacagaact ttgaggtgga ctgcatttaa caatagtcac 4260
aatgttaaaa gaacaaaatt cttgagcagt tttttttttc tgttttgttt taaaaaatgt 4320
tcaggtttat ttgtggaaat gcaagatttc tatgaaaata gtttttgtat ggaaattttt 4380
gtaatacttt ttatcaacaa aataagaaca tgtgttcctg tcaggggtgt gatgtcaagc 4440
atgaacggta gtgcgtgtgc accaccaacg tttggtgaaa actattttta tcaagaaaaa 4500
ggaatcatag aagagaaata ttttcaagtt agatactata aaagctaggt gcactaccac 4560
cacggcttgt cacgccacac ccctgagtcc cacaaggcag ataacatatt gtaatgaaca 4620
gttgtgtgta aaatgataaa agacacagac ctcttgacaa cattgtgaaa acagttgagt 4680
gcacacagtt tgctgtttga atccaatgca caaaaatttt acaaaactcc attaaaatta 4740
tgtccatttt actttcagct ttggctttga tttttctctt gcatgtgtaa atgaatgtaa 4800
catggtggtt ttgtatagaa aatatacatc aaggggtctt aggatctcaa agttagaatc 4860
ttcccaactt acagcaaaaa ggaaaaggcc atcctggagg tgctcctctc tttctctctc 4920
ccttcctctg tctctctctt tgactctgtc tctttgtgtc tcagtctttc tctatatcag 4980
tttttctctg tccctctcta cctctggctc tatcactctc tgtctcattt acacacatac 5040
acacacacac acacacacac acacacacac gaataaagac atatacattg gttttagaat 5100
tagggtagct ggaataaaaa gaatatgatt gtagagatgg caacctttat cttatctcat 5160
ttgtagctgg aaattgacta agttcactgt gctgcattat gttgtggaat ggtaattatc 5220
tactttggtt caactcatat ccaatttcag aattttctgt gcattgatac ttcaataatc 5280
atcagcagag gaacaaaaag ggaaaagttt agaattaata attaatttta gatcctaaca 5340
tattaataga aacaaactat aacagtttta cgttttgaaa atcaaatctg taagattcaa 5400
cttattttcc tgattaatta attaattaat tctaagtgtg caattataat tggaatctga 5460
caaaaaaaaa accccactgg aaaagtttcc ataatgtatt tcttaaaata gtaaaaattg 5520
cataatcaaa ttatctcaaa ttaattaaga tttatatatg tgagcacttt aaatatttta 5580
tgctatgatt attatcagat ttcaatgatt attttgttca agctacaaat gtagctatac 5640
aaatcactgc taaagtagca gtactggtgt attagtgcca accaagttta aataaggaaa 5700
ataatataag tattcagatt attaagatgc tgtttttaac aaacaaattt taaattttat 5760
gaaaatatga atttgtaaag gaaacaaact tcattattaa tattatgggg aaattctgtg 5820
aatatataca atctgacaca tgtagaattt gcacattcga tgagaaactg tgtcaaaaat 5880
gatcaattga agcaactcat ttaataaaaa agaactctca ctaagcaatg ctttaatatg 5940
ttttaaagat ataattttaa acacttggaa atcttatata tgtgagtata aaaacacatt 6000
aaatattcat atcttaattt acttaaaaga gttttaactt caatttatta ctgaatttaa 6060
taatcataaa cattgggtat aaaattacaa ctttattgtt aagcagccag aagagaatga 6120
attctggtaa aatttgataa tgattttatt gaaaaattga attaagttct taaatatgta 6180
acaaccttac tagacattaa ctttataaat gatattaaat gtttataata tattctttta 6240
cattctaatt ctaattaata cttcatacat gtataagtac ttaaattttc caaaacacct 6300
ggtcataata tatatattaa ttttaattat aataaaaatg ctaattactt tcatacataa 6360
tatcaaacaa tgaaaaaact ctaggttgga agcatgtaag agttctcagc attttctaga 6420
ggaaaaatca aaagcaaaga gaagctaata cctgttctag gcaacatcag aacctatgaa 6480
ttgagaggat gaatggaggc tcatggactt cagctataga aaccaacaag gatggagaca 6540
ccagaggttt tatacccaca tttactacca ctaaataatc agtatctccc tgggaacacc 6600
aaagaaacac acataattac cactgaacaa ccctaactct catgggaaca caagagaaat 6660
atagtctagt ttataaatgt gtaaaatgaa gaatagttta ttgatgggaa tatttcagat 6720
attttgttgc tcacttatat ctacatataa gtgcatatgt aataaaaatt catatcccat 6780
ggcaccattc ttgggtggta tagatatgaa acattgctgg tttcaaaatg tttaaggatt 6840
gtgtcaactt ttgatgtctg tttactttga aatatattgt aactgcagaa aagtgcatat 6900
acaaggcata tatacacaga attgctgcta gttgttatag ttttaaacac agttattgtg 6960
ccatttaacc caggctcaat gaaagttctc tgacttgctg ataacatttt cagggaagaa 7020
attacagtga tctttgatca tgaaatgtat aattaactca tctgtgtttg actatgccaa 7080
tagagtgtgt acagcattgt agagaaacag tcttgggatt ttgtggcagc ttttaaaggt 7140
agaatcatag caatgaaaac tttggttatg cctgtcctac ctaaatgtat acaaaatttg 7200
aaccattttc ctccattgtg agctgatacg tgaccagatc tgcagacatg taaaattgtg 7260
aagttattcc cttgtcttta ggtatttgcc tcaatgcaaa ttcaaatgta ataatttttt 7320
ttatttctct taaaatatgt cagttaatta tttgattttt tctcaatttt ggtggtttta 7380
tagaaatgct gaataggctc aatgggtttt agatgtttta tgtctattat tctactctag 7440
tctataatta tctggatatt cattgactcc tagctcaaaa ttagttcctt ggaaaatttg 7500
aagactttcc actatttcct cttccccctc atctatttcc cattttctcc cccatttcca 7560
tctctttccc ccattcctca tcctttatat atatatatat atatatatat acatatatat 7620
atatatatat gtgtgtgtgt gtgtgtgtgt gtgtgtgtgt gtgtgtgtgt gtgtacttgc 7680
ttttttatta tcttgtctaa tgttgagatt gctgtggaaa acctaaaatc atgatcagat 7740
ggtcacagtt ctttgatcat cttcttcttc ttcttcttct tcttcttctt cttcttcttc 7800
ttcttcttct tcttcttctt cttcttcttc ttcttcttcc tcttcctcct cctcctcctc 7860
ttcctcctcc tcttcctctt cgtcgtcttc tccttctcct ccttcttttc cttctcttcc 7920
tccacctctt cctttcttct tcttttccct cttcttcctc cttctcttgt gcctcctctt 7980
tttgtttttc ttacttctct ctttgtctct atctctatgt ttctgtttct ttgtctctgt 8040
ctctctctct gtgtctctgt ctgtcagtct ctgtcggcct gtctgtcttt ctctctgtct 8100
cagatgtaga ttgatctgta tcaattgtac taacccttac tagactcctt aataaagcta 8160
ctatttttca attttaagaa aagaccttga aataatactt cattgtcttc ctttcttcat 8220
atttcctttg ttctctctaa atctgttctt atgacttgta attacttaac tttgaatctt 8280
cagtctattt acttttctat ttctctatat tgttatttta agtctataat ctaaaatact 8340
tttttagttt ctcctatctt tgacattgtt ttaatttctg taagttactg tttaattcct 8400
atgacatctt cacttatatc atagtagttg atcataattg ttctttattc ccatcattag 8460
gaaaagacat tgtatattcc tgttcttttc tatttttttc acctgagttg aaggtattat 8520
tgagataaca gttgagtcat ttaccctcca gttactcctc ccagttcttt tcactttctc 8580
tcccacatgt atctacttcc tgtttgtctc tcattcaaaa aggaaaggcc tctaagagat 8640
aacagcaaaa tttaacaaca taaaatataa taacataaaa cacacaagca aaaactgtca 8700
catcaaagtt gaacaagata aaccaacaaa acaaaaagag cctcaagaag gcagaagaat 8760
cagagagcct cttgttcact tacctcagtt gaattagaat tggttcttat cctgtgggta 8820
gtgacccatt tagaggtaga atgacccttt cttaaagttt tcctaagaca atcagacaat 8880
atagatgttt acattatgat ttataaatag caaaattata gttatgaaat agaggcaaaa 8940
ataattttat ggttttgggt cattacaaca ttaagaacca tatttgaggg ttctagcctt 9000
aggaaggttg aaaaccactg aattataaag ggccttgtct ccttgtgtcc tctacccttt 9060
tgaatctttc acatttctca cctcatcttc tgtgggtggg atttagtgga gacatcccac 9120
tgaaagctgt gttccaaatg catctgtttg ttaaatgtca tccgtcaact cttactgttc 9180
tctttatgat gatgtaaagt agaataaaca tcactggcaa atttttgttt atgttaagtt 9240
gtgacagaat gttcatctaa cttgagtaat cctgtaggtg aatatatgtg gactacttga 9300
tgaataatct gtccaagtac ctccgtttgg tttttacttc ttgggtatat tagatttgca 9360
gagaggtgtc ttttaacttc attcctaatg ccttgaaatc ctgatcacta tatttcagcc 9420
accagagtca agaacttatc actgcaaatt ctgccctgtg tgtgagtgaa taaagcaatt 9480
tttgtcccat ttattctgaa aatttgcgtt taattatatc tcacattctt catactagaa 9540
tatacatttt cctcttttaa aaatatgcac acctgacaga ctgtgtttga aatagtggga 9600
acaccatact ctatggttcc agtgtctgtt ttgaccatca cattactatc ttctctccca 9660
agggccaagt ctaccaagtg gaatatgcct ttaatgctat taaccaggct ggtcttatac 9720
ttgtagctgt cagaggaaaa gactgctcag gttttgtcac atagatgaaa gtacatgaca 9780
aattactaga ttccaacaca cttgttgaag ataactgaaa ccagtggttg tataatgatg 9840
ggagtaacag ctgacagact ccaggtacaa atggcacact atgaggtagt taattggaaa 9900
gacaaaggta actatgagtt tcctgtggac atgctatgta aaagaattgc tgatattact 9960
caagtcaaca cacagaataa tgaaatgagg ctgcttcatt gttgtttgat ttttttttac 10020
tggtataaat gaaaaaacaa ggccctcaag tgtactagtg gaatcctata ggtgttaagc 10080
catgacagca gaagttaaat gaatataacc aaccagcttc cttgaaaaca ataagtgagg 10140
aagaaaattg attggatatt tgaataaaca tttagataat gtaattacat gtctgcctac 10200
tattctgtcg attggtttca aactttcaga aatagaaatt ggaagagtta gagttgaaaa 10260
tactcgattc aggattctta cagaagcaga gattggcact catcttgttg ttctagcaga 10320
gagactgaac attgtcatca gtttaccaaa tctgtgatgc cacttgcctg tgtgtttgat 10380
aaaaaccaac atcatagagg ctccacagct taaaatggaa cctcttccac tcctgccact 10440
gagctgctta ggactctgta taaataaaaa cagtcctttt ggaaaaataa atatgtacac 10500
tgtacttaaa aataaacaca tgaaattttt atgtgctacg ttaaaaccta actccaaaat 10560
ttaaagaaac gctacaattg tccacatcca ttaaaagact ccttgttatt ttatgttctc 10620
ttttgtacca ctattaaatt gattctctat tgcta 10655
<210> 121
<211> 10557
<212> DNA
<213> Mus musculus
<400> 121
aaacaggact aatgcttcta agatgatttt ggatttgggg gttggaggat attagaaact 60
accctctggt aacgaaacga acagattacc gtttaacatt tcttcatctt catactgctc 120
gcgcggctgg acattgtgct tgctggattt ttcccggatg ctcccagact aacatggatg 180
tcccaccatc ccttgcagtg gaagcttgct ccttggcgca gtgagtgaag aacatgcaga 240
gactgctaat gggtttggga agcggagact ccttcctctt tctgtgacca tgccgtgatt 300
gtgtttgcgg ccactattcc acgcatcctt cttctcgtcc aagcccggag cctaacgcta 360
gatcggggaa gtgggtgccg cgcgcgcagg cacggaaaca tcatgaagat tatttcccca 420
gttttaagta atctagtctt cagtcgctcc attaaagtcc tgctctgctt gttgtggatc 480
ggatattcgc aaggaaccac acatgtgtta agattcggtg gtatatttga atatgtggaa 540
tctggcccta tgggagctga agaacttgca ttcagatttg ctgtgaatac aatcaacagg 600
aacaggactc tgctacccaa taccacgtta acatatgata cacagaagat caatctctat 660
gacagttttg aagcatctaa gaaagcttgt gatcagctgt ctcttggggt ggctgccatc 720
ttcggtcctt cacacagttc atcagcaaat gctgttcagt ccatctgcaa tgctctgggg 780
gttcctcaca tacagacccg ctggaagcac caggtgtcag acaataagga ttccttctat 840
gtcagtctct acccagactt ctcttccctc agccgtgcca tcttggattt ggtgcagttt 900
tttaagtgga aaactgtcac agttgtgtat gacgacagca ctggtctcat tcgcttgcaa 960
gagctcatca aagctccatc aaggtacaat cttcgactta aaattcgtca gctgccagct 1020
gatacaaaag atgcaaagcc tttgctgaaa gagatgaaga ggggcaagga gttccacgtg 1080
atcttcgact gcagccatga aatggcagca ggcattttaa agcaggcatt agctatggga 1140
atgatgacag aatactacca ctatatattt acgactctgg acctcttcgc tcttgatgtg 1200
gagccctaca gatacagtgg cgtaaatatg acagggttca gaatactaaa tacagagaat 1260
acccaagtct cctccatcat cgagaagtgg tcgatggaac ggttacaggc acctccaaaa 1320
cctgactcag gtttgctgga tggatttatg acgactgatg ctgctctgat gtatgatgca 1380
gtgcacgttg tgtctgtagc tgtccaacag tttccccaga tgacagtcag ctccttgcaa 1440
tgcaatcgac acaaaccctg gcgctttggg actcgcttca tgagcctaat taaagaggct 1500
cattgggaag gtcttacagg cagaattaca tttaacaaaa ccaatggatt gcgaacagat 1560
tttgatttgg atgtgatcag tctcaaggaa gaaggtctgg agaagattgg gacttgggat 1620
ccatccagtg gcctgaatat gacagaaagt cagaaaggga agccagcaaa tattacagat 1680
tcattgtcta atcgttcttt gattgttacc accattttgg aagaaccata tgtcctgttt 1740
aagaagtctg acaaacctct ctatgggaat gatcgatttg aaggctactg tattgatctt 1800
ctacgagagt tatctacaat ccttggcttt acatatgaaa ttaggcttgt ggaggatggg 1860
aaatatggag cccaggatga tgtgaatgga caatggaatg gaatggttcg tgagctaatt 1920
gatcataaag ctgaccttgc agttgctcca ctggctatta cctatgttcg tgagaaggtc 1980
atcgactttt caaagccgtt tatgactctt ggaataagta ttttgtaccg caagcccaat 2040
ggtacaaacc caggcgtctt ctccttcctg aatcctctct cccctgatat ctggatgtat 2100
attctgctgg cttacttggg tgtcagttgt gtgctctttg tcatagccag gtttagtccc 2160
tatgagtggt ataatccaca cccttgcaac cctgactcag acgtggtgga aaacaatttt 2220
accttgctaa atagtttctg gtttggagtt ggagctctca tgcagcaagg ttctgagctc 2280
atgcccaaag cactctccac caggatagtg ggaggcattt ggtggttttt cacacttatc 2340
atcatttctt cgtataccgc taacctagcc gcctttctga ccgtggaacg catggagtcg 2400
cctattgact ctgctgacga tttagctaag caaaccaaga tagaatatgg agcagtagag 2460
gacggcgcaa ccatgacgtt tttcaagaaa tcaaaaatct caacgtatga taaaatgtgg 2520
gcatttatga gcagcaggag acagtctgtg cttgtcaaaa gcaatgagga agggattcaa 2580
cgtgtcctca cctccgatta tgctttctta atggagtcaa cgaccatcga gtttgttacc 2640
cagcggaact gtaacctcac gcagattggt ggccttatag actccaaagg ctatggtgtt 2700
ggcactccca tgggttctcc atatcgagac aaaatcacca tagccattct tcagctgcag 2760
gaggaaggca agctgcacat gatgaaggag aagtggtggc gaggcaatgg ctgcccagag 2820
gaggagagca aagaggccag tgctctaggg gtgcagaata ttggtggtat cttcattgtc 2880
ctggcagccg gcttggtgct ctcagttttt gtggcagtgg gagagttttt atacaaatcc 2940
aaaaaaaacg ctcaattgga aaagaggtcc ttctgtagcg ccatggtgga agaactgaga 3000
atgtctctga agtgccagcg tcggctcaaa cataagccac aggccccagt tattgtgaaa 3060
acagaagaag ttatcaacat gcacacattt aacgacagaa ggttgccagg taaagaaacc 3120
atggcatgaa gctgggaggc caatcaccca agcacaaact gtcgtctttt tttttttttt 3180
caaacaattt agcgagaatg tttcctgtgg aaatatgcaa cctgtgcaaa ataaaatgag 3240
ttacctcatg ccgctgtgtc tatgaactag agactcttgt gatctaagca gtttcagtga 3300
tcagacttga tttacaagca ccatggatcg acaaagttac acggggttac actgtttatc 3360
atgggttcct cccttccttt gagtgaatgt tacatgaaaa tgttgtggct ggtttcaaat 3420
gcagtccaga gagaaactgc tggttccttc tgaagctcaa ctgttgtcag gagatggaat 3480
gttggggccc aaaaggataa ccaataaaaa tgccataatt tataaaagca aaacaaaaag 3540
cgtgtgaaat ctgcaaaaat tgtagtgtca caagaaacag tatagtccca tggtcaccaa 3600
caaaatgagg tgataatgtt actagccccc aatactcagt aaaatcatca tctgaataga 3660
taatgtgttc atagaatgtg gaaaaaatgt aatgcaaaac atatcagtat tcaatcaaag 3720
tggaacagaa agcagaccac catcagttat tttcctttct caatagtctg tgtcatggat 3780
tgtgatatag atggcaatta tctatctaat tgttttctta aaatacccat ggcaaatatt 3840
ttaaaatgca acttgctccc aggaacccct accctaacct acactagaaa taaaaaagcc 3900
accactggta taaagattct gatgtaaaag atatgttttt caatccttgt catgaattgt 3960
aaaacagggc tcagtattac tggttatatg gaagactgaa gctttcactc tgacattctg 4020
atatgtcagc tgaaactctc cttcctcctg gaaaggacct tgatggagcc tgggcagatt 4080
ccattgataa gactggggac ttgtcaccta tacagaacta cgtgacagaa ctttgaggtg 4140
gactgcattt aacaatagtc acaatgttaa aagaacaaaa ttcttgagca gttttttttt 4200
tctgttttgt tttaaaaaat gttcaggttt atttgtggaa atgcaagatt tctatgaaaa 4260
tagtttttgt atggaaattt ttgtaatact ttttatcaac aaaataagaa catgtgttcc 4320
tgtcaggggt gtgatgtcaa gcatgaacgg tagtgcgtgt gcaccaccaa cgtttggtga 4380
aaactatttt tatcaagaaa aaggaatcat agaagagaaa tattttcaag ttagatacta 4440
taaaagctag gtgcactacc accacggctt gtcacgccac acccctgagt cccacaaggc 4500
agataacata ttgtaatgaa cagttgtgtg taaaatgata aaagacacag acctcttgac 4560
aacattgtga aaacagttga gtgcacacag tttgctgttt gaatccaatg cacaaaaatt 4620
ttacaaaact ccattaaaat tatgtccatt ttactttcag ctttggcttt gatttttctc 4680
ttgcatgtgt aaatgaatgt aacatggtgg ttttgtatag aaaatataca tcaaggggtc 4740
ttaggatctc aaagttagaa tcttcccaac ttacagcaaa aaggaaaagg ccatcctgga 4800
ggtgctcctc tctttctctc tcccttcctc tgtctctctc tttgactctg tctctttgtg 4860
tctcagtctt tctctatatc agtttttctc tgtccctctc tacctctggc tctatcactc 4920
tctgtctcat ttacacacat acacacacac acacacacac acacacacac acgaataaag 4980
acatatacat tggttttaga attagggtag ctggaataaa aagaatatga ttgtagagat 5040
ggcaaccttt atcttatctc atttgtagct ggaaattgac taagttcact gtgctgcatt 5100
atgttgtgga atggtaatta tctactttgg ttcaactcat atccaatttc agaattttct 5160
gtgcattgat acttcaataa tcatcagcag aggaacaaaa agggaaaagt ttagaattaa 5220
taattaattt tagatcctaa catattaata gaaacaaact ataacagttt tacgttttga 5280
aaatcaaatc tgtaagattc aacttatttt cctgattaat taattaatta attctaagtg 5340
tgcaattata attggaatct gacaaaaaaa aaaccccact ggaaaagttt ccataatgta 5400
tttcttaaaa tagtaaaaat tgcataatca aattatctca aattaattaa gatttatata 5460
tgtgagcact ttaaatattt tatgctatga ttattatcag atttcaatga ttattttgtt 5520
caagctacaa atgtagctat acaaatcact gctaaagtag cagtactggt gtattagtgc 5580
caaccaagtt taaataagga aaataatata agtattcaga ttattaagat gctgttttta 5640
acaaacaaat tttaaatttt atgaaaatat gaatttgtaa aggaaacaaa cttcattatt 5700
aatattatgg ggaaattctg tgaatatata caatctgaca catgtagaat ttgcacattc 5760
gatgagaaac tgtgtcaaaa atgatcaatt gaagcaactc atttaataaa aaagaactct 5820
cactaagcaa tgctttaata tgttttaaag atataatttt aaacacttgg aaatcttata 5880
tatgtgagta taaaaacaca ttaaatattc atatcttaat ttacttaaaa gagttttaac 5940
ttcaatttat tactgaattt aataatcata aacattgggt ataaaattac aactttattg 6000
ttaagcagcc agaagagaat gaattctggt aaaatttgat aatgatttta ttgaaaaatt 6060
gaattaagtt cttaaatatg taacaacctt actagacatt aactttataa atgatattaa 6120
atgtttataa tatattcttt tacattctaa ttctaattaa tacttcatac atgtataagt 6180
acttaaattt tccaaaacac ctggtcataa tatatatatt aattttaatt ataataaaaa 6240
tgctaattac tttcatacat aatatcaaac aatgaaaaaa ctctaggttg gaagcatgta 6300
agagttctca gcattttcta gaggaaaaat caaaagcaaa gagaagctaa tacctgttct 6360
aggcaacatc agaacctatg aattgagagg atgaatggag gctcatggac ttcagctata 6420
gaaaccaaca aggatggaga caccagaggt tttataccca catttactac cactaaataa 6480
tcagtatctc cctgggaaca ccaaagaaac acacataatt accactgaac aaccctaact 6540
ctcatgggaa cacaagagaa atatagtcta gtttataaat gtgtaaaatg aagaatagtt 6600
tattgatggg aatatttcag atattttgtt gctcacttat atctacatat aagtgcatat 6660
gtaataaaaa ttcatatccc atggcaccat tcttgggtgg tatagatatg aaacattgct 6720
ggtttcaaaa tgtttaagga ttgtgtcaac ttttgatgtc tgtttacttt gaaatatatt 6780
gtaactgcag aaaagtgcat atacaaggca tatatacaca gaattgctgc tagttgttat 6840
agttttaaac acagttattg tgccatttaa cccaggctca atgaaagttc tctgacttgc 6900
tgataacatt ttcagggaag aaattacagt gatctttgat catgaaatgt ataattaact 6960
catctgtgtt tgactatgcc aatagagtgt gtacagcatt gtagagaaac agtcttggga 7020
ttttgtggca gcttttaaag gtagaatcat agcaatgaaa actttggtta tgcctgtcct 7080
acctaaatgt atacaaaatt tgaaccattt tcctccattg tgagctgata cgtgaccaga 7140
tctgcagaca tgtaaaattg tgaagttatt cccttgtctt taggtatttg cctcaatgca 7200
aattcaaatg taataatttt ttttatttct cttaaaatat gtcagttaat tatttgattt 7260
tttctcaatt ttggtggttt tatagaaatg ctgaataggc tcaatgggtt ttagatgttt 7320
tatgtctatt attctactct agtctataat tatctggata ttcattgact cctagctcaa 7380
aattagttcc ttggaaaatt tgaagacttt ccactatttc ctcttccccc tcatctattt 7440
cccattttct cccccatttc catctctttc ccccattcct catcctttat atatatatat 7500
atatatatat atacatatat atatatatat atgtgtgtgt gtgtgtgtgt gtgtgtgtgt 7560
gtgtgtgtgt gtgtgtactt gcttttttat tatcttgtct aatgttgaga ttgctgtgga 7620
aaacctaaaa tcatgatcag atggtcacag ttctttgatc atcttcttct tcttcttctt 7680
cttcttcttc ttcttcttct tcttcttctt cttcttcttc ttcttcttct tcttcttctt 7740
cctcttcctc ctcctcctcc tcttcctcct cctcttcctc ttcgtcgtct tctccttctc 7800
ctccttcttt tccttctctt cctccacctc ttcctttctt cttcttttcc ctcttcttcc 7860
tccttctctt gtgcctcctc tttttgtttt tcttacttct ctctttgtct ctatctctat 7920
gtttctgttt ctttgtctct gtctctctct ctgtgtctct gtctgtcagt ctctgtcggc 7980
ctgtctgtct ttctctctgt ctcagatgta gattgatctg tatcaattgt actaaccctt 8040
actagactcc ttaataaagc tactattttt caattttaag aaaagacctt gaaataatac 8100
ttcattgtct tcctttcttc atatttcctt tgttctctct aaatctgttc ttatgacttg 8160
taattactta actttgaatc ttcagtctat ttacttttct atttctctat attgttattt 8220
taagtctata atctaaaata cttttttagt ttctcctatc tttgacattg ttttaatttc 8280
tgtaagttac tgtttaattc ctatgacatc ttcacttata tcatagtagt tgatcataat 8340
tgttctttat tcccatcatt aggaaaagac attgtatatt cctgttcttt tctatttttt 8400
tcacctgagt tgaaggtatt attgagataa cagttgagtc atttaccctc cagttactcc 8460
tcccagttct tttcactttc tctcccacat gtatctactt cctgtttgtc tctcattcaa 8520
aaaggaaagg cctctaagag ataacagcaa aatttaacaa cataaaatat aataacataa 8580
aacacacaag caaaaactgt cacatcaaag ttgaacaaga taaaccaaca aaacaaaaag 8640
agcctcaaga aggcagaaga atcagagagc ctcttgttca cttacctcag ttgaattaga 8700
attggttctt atcctgtggg tagtgaccca tttagaggta gaatgaccct ttcttaaagt 8760
tttcctaaga caatcagaca atatagatgt ttacattatg atttataaat agcaaaatta 8820
tagttatgaa atagaggcaa aaataatttt atggttttgg gtcattacaa cattaagaac 8880
catatttgag ggttctagcc ttaggaaggt tgaaaaccac tgaattataa agggccttgt 8940
ctccttgtgt cctctaccct tttgaatctt tcacatttct cacctcatct tctgtgggtg 9000
ggatttagtg gagacatccc actgaaagct gtgttccaaa tgcatctgtt tgttaaatgt 9060
catccgtcaa ctcttactgt tctctttatg atgatgtaaa gtagaataaa catcactggc 9120
aaatttttgt ttatgttaag ttgtgacaga atgttcatct aacttgagta atcctgtagg 9180
tgaatatatg tggactactt gatgaataat ctgtccaagt acctccgttt ggtttttact 9240
tcttgggtat attagatttg cagagaggtg tcttttaact tcattcctaa tgccttgaaa 9300
tcctgatcac tatatttcag ccaccagagt caagaactta tcactgcaaa ttctgccctg 9360
tgtgtgagtg aataaagcaa tttttgtccc atttattctg aaaatttgcg tttaattata 9420
tctcacattc ttcatactag aatatacatt ttcctctttt aaaaatatgc acacctgaca 9480
gactgtgttt gaaatagtgg gaacaccata ctctatggtt ccagtgtctg ttttgaccat 9540
cacattacta tcttctctcc caagggccaa gtctaccaag tggaatatgc ctttaatgct 9600
attaaccagg ctggtcttat acttgtagct gtcagaggaa aagactgctc aggttttgtc 9660
acatagatga aagtacatga caaattacta gattccaaca cacttgttga agataactga 9720
aaccagtggt tgtataatga tgggagtaac agctgacaga ctccaggtac aaatggcaca 9780
ctatgaggta gttaattgga aagacaaagg taactatgag tttcctgtgg acatgctatg 9840
taaaagaatt gctgatatta ctcaagtcaa cacacagaat aatgaaatga ggctgcttca 9900
ttgttgtttg attttttttt actggtataa atgaaaaaac aaggccctca agtgtactag 9960
tggaatccta taggtgttaa gccatgacag cagaagttaa atgaatataa ccaaccagct 10020
tccttgaaaa caataagtga ggaagaaaat tgattggata tttgaataaa catttagata 10080
atgtaattac atgtctgcct actattctgt cgattggttt caaactttca gaaatagaaa 10140
ttggaagagt tagagttgaa aatactcgat tcaggattct tacagaagca gagattggca 10200
ctcatcttgt tgttctagca gagagactga acattgtcat cagtttacca aatctgtgat 10260
gccacttgcc tgtgtgtttg ataaaaacca acatcataga ggctccacag cttaaaatgg 10320
aacctcttcc actcctgcca ctgagctgct taggactctg tataaataaa aacagtcctt 10380
ttggaaaaat aaatatgtac actgtactta aaaataaaca catgaaattt ttatgtgcta 10440
cgttaaaacc taactccaaa atttaaagaa acgctacaat tgtccacatc cattaaaaga 10500
ctccttgtta ttttatgttc tcttttgtac cactattaaa ttgattctct attgcta 10557
<210> 122
<211> 4579
<212> DNA
<213> Macaca mulatta
<400> 122
gctcgcgcgg ccggacattg tgggtgtgcg tgctggattt ctcccggatg ctctccgact 60
aacatggatg tcccaccatt ccttgcagtg gaaggttgtt ccttggcgca gtgagtgaag 120
aacatgcagc gattgctaat gggtttggga agcggagact ccttcctctc tctgtgacca 180
tgccgtgatc gtgtctgcgg tcaccactcg acgcatcctt atttctgccc gaacccggga 240
gccgaacgct agatcgggga agtgggtgcc gtgcgtgtgg gcacagaaac accatgaaga 300
ttattttccc cattctaagt aatccagtct tcaggcgcac cgttaaactc ctgctctgtt 360
tactgtggat tggatattct caaggaacta cacatgtatt aagatttggt ggtatttttg 420
aatatgtgga atctggccca atgggagctg aggaacttgc attcagattt gctgtgaata 480
caattaacag aaacagaaca ttgctaccca atactaccct tacctatgat acccagaaga 540
taaaccttta tgatagtttt gaagcatcca agaaagcctg tgatcagctg tctcttgggg 600
tggctgccat cttcgggcct tcacacagct catcagcaaa tgcagtgcag tccatctgca 660
atgctctggg agttccccac atacagaccc gctggaagca ccaggtgtca gacaacaaag 720
attccttcta tgtcagtctc tacccagact tctcttcact cagccgtgcc attttagacc 780
tggtgcagtt tttcaagtgg aaaaccgtca cggttgtgta tgatgacagc actggtctca 840
ttcgtttgca agagctcatc aaagctccat caaggtacaa tcttcgactc aaaattcgtc 900
agctacctgc tgatacaaag gatgcaaaac ccttactaaa agaaatgaaa agaggcaagg 960
agtttcacgt aatctttgat tgtagccatg aaatggcagc aggcatttta aaacaggcat 1020
tagctatggg aatgatgaca gaatactatc attacatctt taccactctg gacctctttg 1080
ctcttgacgt tgagccgtac cgatacagtg gtgttaatat gacagggttc agaatattaa 1140
atacagaaaa tacccaagtt tcctccatca ttgaaaagtg gtcaatggaa cgattgcagg 1200
cacctccgaa acccgattca ggtttgctgg atggatttat gacgactgat gctgctctaa 1260
tgtatgatgc tgtgcatgtg gtgtctgtgg ccgtccaaca gtttccccag atgacagtca 1320
gttccttgca gtgtaatcga cataaaccct ggcgcttcgg gactcgcttt atgagtctaa 1380
ttaaagaggc acattgggaa ggcctcacag gcagaataac tttcaacaaa accaatggct 1440
tgcgaacaga ttttgatttg gatgtgatca gtctcaagga agaaggtcta gaaaagattg 1500
gaacgtggga tccagccagt ggcctgaata tgacagaaag tcaaaaggga aagccggcaa 1560
acatcacaga ttccttatcc aatcgttctt tgattgttac caccattttg gaagaaccct 1620
atgtcctttt taagaagtct gacaaacctc tctatggtaa tgatcgattt gaaggctatt 1680
gcattgatct cctcagagag ttatctacaa tccttggctt tacatatgaa attagacttg 1740
tggaagatgg gaaatatgga gcccaggatg atgccaatgg acaatggaat ggaatggttc 1800
gtgaactaat tgatcataaa gctgaccttg cagttgctcc actggctatt acctatgttc 1860
gagagaaggt catcgacttt tccaagccct ttatgacact tggaataagt attttgtacc 1920
gcaagcccaa tggtacaaac ccaggcgtct tctccttcct gaatcctctc tcccctgata 1980
tctggatgta tattctgctg gcttacttgg gtgtcagttg tgtgctcttt gtcatagcca 2040
ggtttagtcc ttatgagtgg tataatccac acccttgcaa ccctgactca gacgtggtgg 2100
aaaacaattt taccttgcta aatagtttct ggtttggagt tggagctctc atgcagcaag 2160
gttctgagct catgcccaaa gcactgtcca ccaggatagt gggaggcatt tggtggtttt 2220
tcacacttat catcatttct tcgtatactg ctaacttagc cgcctttctg acagtggaac 2280
gcatggaatc ccctattgac tctgctgatg atttagctaa acaaaccaag atagaatatg 2340
gagcagtaga ggatggtgca accatgactt ttttcaagaa atcaaaaatc tccacgtatg 2400
acaaaatgtg ggcctttatg agtagcagaa ggcagtcagt gctggtcaaa agtaatgaag 2460
aaggaatcca gcgagtcctc acctctgatt atgctttcct aatggagtca acaaccatcg 2520
agtttgttac ccagcggaac tgtaacctga cacagattgg cggccttata gactccaaag 2580
gttatggcgt tggcactccc atgggttctc catatcgaga caaaattacc atagcaattc 2640
ttcagctgca agaggaaggc aagctgcata tgatgaagga gaaatggtgg aggggcaatg 2700
gttgcccaga agaggagagc aaagaggcca gtgccctggg ggttcagaat attggtggca 2760
tcttcattgt tctggcagcc ggcttggtgc tttcagtttt tgtggcagtg ggagaatttt 2820
tatacaaatc caaaaaaaac gctcagttgg aaaagaggtc cttctgtagt gccatggtag 2880
aagaattgag gatgtccctg aagtgccagc gtcggttaaa acataagcca caggccccag 2940
ttattgtgaa aacagaagaa gttatcaaca tgcacacatt taacgacaga aggttgccag 3000
gtaaagaaac catggcataa agctgggagg ccaaacaccc aagcacaaac tgtcgtcttt 3060
ttccaaacaa tttagcgaga atgtttcctg tggaaatatg caacctgtgc aaaataaaat 3120
gagttacctc atgccgctgt gtctatgaac tagagactct gtgatctaag caattgcaat 3180
gatcagactt gatttacaag catcatggat caaccaagtt acacggggtt acactgttaa 3240
tcatgggttc ctcccttctt ctgagtgaat gttacatgag cattttgtgg ctggtttcaa 3300
atgcagtcca gtgagaaatt acaggttcct tttgaagctc aactgttgcc aggagatgga 3360
atatcgacgc ccaacagggc aaccaatcaa agtgtcacta agaatacaaa tatttggaat 3420
cagcaaaaac tgtagtgtta cagaacagta cagtcttctg aacacccaga tcatagaggt 3480
gatgatgcta ctagccccca actactcagt ataattattg tctgaataca cagtatatgt 3540
ttataggatg tgaaaaaatg taatgcaaaa caaatttgaa tcccatggca gttggaatat 3600
aaagcagatg tttatcactt attttccttt ttttcttttc ctttttgttt tttttttttt 3660
tgacagtctg tgtctctgat tgagatagaa atgccaattt ttaaggcaat aatgcttttc 3720
ttaagttccc taaggcagaa gatttaacat gcaactctac catatccctt tctattcccc 3780
caacaccttt tctctaacct ccgtatccca aataataata ataatagtaa taataaaaac 3840
agttggttca gtgattctga attaaaagga taatgttttg caatctttaa gttgtaaaaa 3900
gggctgagta ttggctgtgt ggaagactaa agctttcatt ctaacattca gacatagcaa 3960
tccaaaccct gttcctgctg taaatgaact tgatggagca tgggcagatc tcagtggtac 4020
gagaaagggg actggtcatc tatagaaaaa tctgtgagag aacttggaca tggactgcat 4080
ttatcaatac agtcacaatg ttaaatgaac aaaattcttg aacagttttt tttcaaaaaa 4140
tgttcaggtt tatttgtgga aatgcaagat ttctatgaaa atagtttttg tatggaaatt 4200
tttgtaatac tttttatcaa caaaacaaga acatgtgttc ctgtcagggg tgtgatgtca 4260
agcatgaatg gtagtgcgtg tgcaccacca acgtttggtg aaaactattt ttatcaagag 4320
aaaaggaatc atagaagaga aatattttca agttagataa tataaaagct aggtgcacta 4380
ccaccactgc ttaccatgcc acacccctgg tttccacgag gctgacaaca tactgtaatg 4440
aacaattgtg tgtaaaatgg taaaagacac agacctcttg acaacattgt gataactgtt 4500
gagtgcacac agtttgctgt ttgaatccaa tgcacaaaat taaaaaaaaa tcattaaaat 4560
tatgttcatt ttactttca 4579
<210> 123
<211> 4722
<212> DNA
<213> Macaca mulatta
<400> 123
ccgtcttcgc ctttgccgtc actcgcgctc ggcgccggcg gctgcgctgg tcggtctggg 60
agccggggac tttccgcccg acctcctccg gctgctcctc cccgaggacc accccacccc 120
ctccccaccc acctcacccc tagcgccagg ctcgcgcggc cggacattgt gggtgtgcgt 180
gctggatttc tcccggatgc tctccgacta acatggatgt cccaccattc cttgcagtgg 240
aaggttgttc cttggcgcag tgagtgaaga acatgcagcg attgctaatg ggtttgggaa 300
gcggagactc cttcctctct ctgtgaccat gccgtgatcg tgtctgcggt caccactcga 360
cgcatcctta tttctgcccg aacccgggag ccgaacgcta gatcggggaa gtgggtgccg 420
tgcgtgtggg cacagaaaca ccatgaagat tattttcccc attctaagta atccagtctt 480
caggcgcacc gttaaactcc tgctctgttt actgtggatt ggatattctc aaggaactac 540
acatgtatta agatttggtg gtatttttga atatgtggaa tctggcccaa tgggagctga 600
ggaacttgca ttcagatttg ctgtgaatac aattaacaga aacagaacat tgctacccaa 660
tactaccctt acctatgata cccagaagat aaacctttat gatagttttg aagcatccaa 720
gaaagcctgt gatcagctgt ctcttggggt ggctgccatc ttcgggcctt cacacagctc 780
atcagcaaat gcagtgcagt ccatctgcaa tgctctggga gttccccaca tacagacccg 840
ctggaagcac caggtgtcag acaacaaaga ttccttctat gtcagtctct acccagactt 900
ctcttcactc agccgtgcca ttttagacct ggtgcagttt ttcaagtgga aaaccgtcac 960
ggttgtgtat gatgacagca ctggtctcat tcgtttgcaa gagctcatca aagctccatc 1020
aaggtacaat cttcgactca aaattcgtca gctacctgct gatacaaagg atgcaaaacc 1080
cttactaaaa gaaatgaaaa gaggcaagga gtttcacgta atctttgatt gtagccatga 1140
aatggcagca ggcattttaa aacaggcatt agctatggga atgatgacag aatactatca 1200
ttacatcttt accactctgg acctctttgc tcttgacgtt gagccgtacc gatacagtgg 1260
tgttaatatg acagggttca gaatattaaa tacagaaaat acccaagttt cctccatcat 1320
tgaaaagtgg tcaatggaac gattgcaggc acctccgaaa cccgattcag gtttgctgga 1380
tggatttatg acgactgatg ctgctctaat gtatgatgct gtgcatgtgg tgtctgtggc 1440
cgtccaacag tttccccaga tgacagtcag ttccttgcag tgtaatcgac ataaaccctg 1500
gcgcttcggg actcgcttta tgagtctaat taaagaggca cattgggaag gcctcacagg 1560
cagaataact ttcaacaaaa ccaatggctt gcgaacagat tttgatttgg atgtgatcag 1620
tctcaaggaa gaaggtctag aaaagattgg aacgtgggat ccagccagtg gcctgaatat 1680
gacagaaagt caaaagggaa agccggcaaa catcacagat tccttatcca atcgttcttt 1740
gattgttacc accattttgg aagaacccta tgtccttttt aagaagtctg acaaacctct 1800
ctatggtaat gatcgatttg aaggctattg cattgatctc ctcagagagt tatctacaat 1860
ccttggcttt acatatgaaa ttagacttgt ggaagatggg aaatatggag cccaggatga 1920
tgccaatgga caatggaatg gaatggttcg tgaactaatt gatcataaag ctgaccttgc 1980
agttgctcca ctggctatta cctatgttcg agagaaggtc atcgactttt ccaagccctt 2040
tatgacactt ggaataagta ttttgtaccg caagcccaat ggtacaaacc caggcgtctt 2100
ctccttcctg aatcctctct cccctgatat ctggatgtat attctgctgg cttacttggg 2160
tgtcagttgt gtgctctttg tcatagccag gtttagtcct tatgagtggt ataatccaca 2220
cccttgcaac cctgactcag acgtggtgga aaacaatttt accttgctaa atagtttctg 2280
gtttggagtt ggagctctca tgcagcaagg ttctgagctc atgcccaaag cactgtccac 2340
caggatagtg ggaggcattt ggtggttttt cacacttatc atcatttctt cgtatactgc 2400
taacttagcc gcctttctga cagtggaacg catggaatcc cctattgact ctgctgatga 2460
tttagctaaa caaaccaaga tagaatatgg agcagtagag gatggtgcaa ccatgacttt 2520
tttcaagaaa tcaaaaatct ccacgtatga caaaatgtgg gcctttatga gtagcagaag 2580
gcagtcagtg ctggtcaaaa gtaatgaaga aggaatccag cgagtcctca cctctgatta 2640
tgctttccta atggagtcaa caaccatcga gtttgttacc cagcggaact gtaacctgac 2700
acagattggc ggccttatag actccaaagg ttatggcgtt ggcactccca tgggttctcc 2760
atatcgagac aaaattacca tagcaattct tcagctgcaa gaggaaggca agctgcatat 2820
gatgaaggag aaatggtgga ggggcaatgg ttgcccagaa gaggagagca aagaggccag 2880
tgccctgggg gttcagaata ttggtggcat cttcattgtt ctggcagccg gcttggtgct 2940
ttcagttttt gtggcagtgg gagaattttt atacaaatcc aaaaaaaacg ctcagttgga 3000
aaagaggtcc ttctgtagtg ccatggtaga agaattgagg atgtccctga agtgccagcg 3060
tcggttaaaa cataagccac aggccccagt tattgtgaaa acagaagaag ttatcaacat 3120
gcacacattt aacgacagaa ggttgccagg taaagaaacc atggcataaa gctgggaggc 3180
caaacaccca agcacaaact gtcgtctttt tccaaacaat ttagcgagaa tgtttcctgt 3240
ggaaatatgc aacctgtgca aaataaaatg agttacctca tgccgctgtg tctatgaact 3300
agagactctg tgatctaagc aattgcaatg atcagacttg atttacaagc atcatggatc 3360
aaccaagtta cacggggtta cactgttaat catgggttcc tcccttcttc tgagtgaatg 3420
ttacatgagc attttgtggc tggtttcaaa tgcagtccag tgagaaatta caggttcctt 3480
ttgaagctca actgttgcca ggagatggaa tatcgacgcc caacagggca accaatcaaa 3540
gtgtcactaa gaatacaaat atttggaatc agcaaaaact gtagtgttac agaacagtac 3600
agtcttctga acacccagat catagaggtg atgatgctac tagcccccaa ctactcagta 3660
taattattgt ctgaatacac agtatatgtt tataggatgt gaaaaaatgt aatgcaaaac 3720
aaatttgaat cccatggcag ttggaatata aagcagatgt ttatcactta ttttcctttt 3780
tttcttttcc tttttgtttt tttttttttt gacagtctgt gtctctgatt gagatagaaa 3840
tgccaatttt taaggcaata atgcttttct taagttccct aaggcagaag atttaacatg 3900
caactctacc atatcccttt ctattccccc aacacctttt ctctaacctc cgtatcccaa 3960
ataataataa taatagtaat aataaaaaca gttggttcag tgattctgaa ttaaaaggat 4020
aatgttttgc aatctttaag ttgtaaaaag ggctgagtat tggctgtgtg gaagactaaa 4080
gctttcattc taacattcag acatagcaat ccaaaccctg ttcctgctgt aaatgaactt 4140
gatggagcat gggcagatct cagtggtacg agaaagggga ctggtcatct atagaaaaat 4200
ctgtgagaga acttggacat ggactgcatt tatcaataca gtcacaatgt taaatgaaca 4260
aaattcttga acagtttttt ttcaaaaaat gttcaggttt atttgtggaa atgcaagatt 4320
tctatgaaaa tagtttttgt atggaaattt ttgtaatact ttttatcaac aaaacaagaa 4380
catgtgttcc tgtcaggggt gtgatgtcaa gcatgaatgg tagtgcgtgt gcaccaccaa 4440
cgtttggtga aaactatttt tatcaagaga aaaggaatca tagaagagaa atattttcaa 4500
gttagataat ataaaagcta ggtgcactac caccactgct taccatgcca cacccctggt 4560
ttccacgagg ctgacaacat actgtaatga acaattgtgt gtaaaatggt aaaagacaca 4620
gacctcttga caacattgtg ataactgttg agtgcacaca gtttgctgtt tgaatccaat 4680
gcacaaaatt aaaaaaaaat cattaaaatt atgttcattt ta 4722
<210> 124
<211> 3058
<212> DNA
<213> Rattus norvegicus
<400> 124
ggctggacat tgtgcttgct ggatttttcc cggatgctcc cggactaaca tggatgtccc 60
accatccctt gcagtggaag cttgctcctt ggcgcagtga gagtgaagaa catgcagcga 120
ctgctaatgg gtttgggaag cggagactcc ttcctctttc tgtgaccatg ccgtgattgt 180
gtctgcggcc actactccac gcatcttcct tctcgtccaa gcccggagcc taacgctaga 240
tcggggaagt gggtgccgcg cgcgcaggca cggaaacatc atgaagatta tttccccagt 300
tttaagtaat ctagtcttca gtcgctccat taaagtcctg ctctgcttat tgtggatcgg 360
atattcgcaa ggaaccacac atgtgttaag attcggtggt atatttgaat atgtggaatc 420
tggccccatg ggagcagaag aacttgcatt cagatttgct gtgaatacca tcaacagaaa 480
caggactttg ctgcccaaca ccactttaac ttatgatact cagaagatca atctctatga 540
cagttttgaa gcatctaaga aagcttgtga tcagctgtct cttggggtgg ctgctatctt 600
cggtccttca cacagttcat cagccaatgc tgtgcagtcc atctgcaatg ctctgggggt 660
tccccacata cagacccgct ggaagcacca ggtgtcagac aacaaggatt ccttctacgt 720
cagtctctac ccagacttct cttccctgag ccgcgccatc ttggatttgg tgcagttttt 780
taagtggaaa actgtcacag ttgtgtatga cgacagcact ggtctcattc gcttgcaaga 840
gctcatcaaa gctccatcga ggtacaatct tcgacttaaa attcgtcagc tgccagctga 900
taccaaagat gcaaaacctt tgctgaagga gatgaaaaga ggcaaggagt tccacgtgat 960
cttcgactgc agccatgaga tggcagcagg cattttaaaa caggcattag ctatgggaat 1020
gatgacagaa tactatcact atatatttac aactctggac ctctttgctc ttgacgtgga 1080
gccctacaga tacagtggcg taaatatgac agggttcagg atactaaata cagagaatac 1140
ccaagtctcc tccatcatcg aaaagtggtc tatggaacgg ttacaggcgc ctccaaaacc 1200
tgactcaggt ttgctggatg gatttatgac gactgatgct gctctgatgt atgatgcagt 1260
gcacgttgtg tctgtggctg tccaacagtt tccccagatg acagtcagct ccttgcaatg 1320
caatcgacac aaaccctggc gctttgggac ccgcttcatg agtctaatta aagaggctca 1380
ctgggaaggt ctcacaggca gaataacatt taacaaaacc aatggattac ggacagattt 1440
tgatttggat gtgatcagtc tcaaggaaga aggtctggag aagattggaa cttgggatcc 1500
agccagtggc ctgaatatga cagaaagtca gaaaggaaag ccagcaaata tcacagactc 1560
attgtctaat cgttctttga ttgttaccac cattttggaa gaaccgtatg ttctgtttaa 1620
gaagtctgac aaaccactct atgggaatga tcgatttgaa ggctactgta ttgatctcct 1680
acgagagtta tctacaatcc ttggctttac atatgagatt aggcttgtgg aggatgggaa 1740
atatggagcc caggatgatg tgaacggaca atggaatgga atggttcgtg aactaatcga 1800
tcataaagct gaccttgcag ttgctccact ggctataacc tatgttcgtg agaaggtcat 1860
cgacttttca aagccgttta tgacacttgg aataagtatt ttgtaccgca agcccaatgg 1920
tacaaaccca ggcgtcttct ccttcctgaa tcctctctcc cctgatatct ggatgtatgt 1980
tctgctggct tgcttgggtg tcagttgtgt gctctttgtc atagccaggt ttagtcccta 2040
tgagtggtat aacccacacc cttgcaaccc tgactcagac gtggtggaaa acaattttac 2100
cttgctaaat agtttctggt ttggagttgg agctctcatg cggcaaggtt ctgagctcat 2160
gcccaaagca ctctccacca ggatagtggg aggcatttgg tggtttttca cacttatcat 2220
catttcttcg tataccgcta acctagccgc ctttctgact gtggaacgca tggagtcgcc 2280
cattgactct gctgacgatt tagctaagca aaccaagata gagtatggag cagtggagga 2340
cggcgcaacc atgacgtttt ttaagaaatc aaaaatttca acgtatgata aaatgtgggc 2400
gtttatgagc agcaggagac agtctgtgct tgtcaaaagc aatgaggaag ggatccaacg 2460
agtcctcacc tcggattatg ctttcttaat ggagtcaaca accatcgagt ttgttacaca 2520
gcggaactgt aacctcacgc agattggcgg ccttatagac tccaaaggct atggcgttgg 2580
cactcctatg ggctctccat atcgagacaa aatcaccata gcaattcttc agctgcagga 2640
ggaaggcaag ctgcacatga tgaaggagaa atggtggcgg ggcaatggct gcccagagga 2700
ggagagcaaa gaggccagtg ctctgggggt gcagaatatt ggtggtatct tcattgtcct 2760
ggcagccggc ttggtgctct cagtttttgt ggcagtggga gagtttttat acaaatccaa 2820
aaaaaacgct caattggaaa agaggtcctt ctgtagcgct atggtggaag agctgagaat 2880
gtccctgaag tgccagcgtc ggctcaaaca taagccacag gccccagtta ttgtgaaaac 2940
agaagaagtt atcaacatgc acacatttaa cgacagaagg ttgccaggta aagaaaccat 3000
ggcatgaagc tgggaggcca atcacccaag cacaaactgt cgtctttttt tttttttt 3058
<210> 125
<211> 2631
<212> DNA
<213> Homo sapiens
<400> 125
accacacatg tattaagatt tggtggtatt tttgaatatg tggaatctgg cccaatggga 60
gctgaggaac ttgcattcag atttgctgtg aacacaatta acagaaacag aacattgcta 120
cccaatacta cccttaccta tgatacccag aagataaacc tttatgatag ttttgaagca 180
tccaagaaag cctgtgatca gctgtctctt ggggtggctg ccatcttcgg gccttcacac 240
agctcatcag caaacgcagt gcagtccatc tgcaatgctc tgggagttcc ccacatacag 300
acccgctgga agcaccaggt gtcagacaac aaagattcct tctatgtcag tctctaccca 360
gacttctctt cactcagccg tgccatttta gacctggtgc agtttttcaa gtggaaaacc 420
gtcacggttg tgtatgatga cagcactggt ctcattcgtt tgcaagagct catcaaagct 480
ccatcaaggt ataatcttcg actcaaaatt cgtcagttac ctgctgatac aaaggatgca 540
aaacccttac taaaagaaat gaaaagaggc aaggagtttc atgtaatctt tgattgtagc 600
catgaaatgg cagcaggcat tttaaaacag gcattagcta tgggaatgat gacagaatac 660
tatcattata tctttaccac tctggacctc tttgctcttg atgttgagcc ctaccgatac 720
agtggtgtta acatgacagg gttcagaata ttaaatacag aaaataccca agtctcctcc 780
atcattgaaa agtggtcgat ggaacgattg caggcacctc cgaaacccga ttcaggtttg 840
ctggatggat ttatgacgac tgatgctgct ctaatgtatg atgctgtgca tgtggtgtct 900
gtggccgttc aacagtttcc ccagatgaca gtcagttcct tgcagtgtaa tcgacataaa 960
ccctggcgct tcgggacccg ctttatgagt ctaattaaag aggcacattg ggaaggcctc 1020
acaggcagaa taactttcaa caaaaccaat ggcttgagaa cagattttga tttggatgtg 1080
atcagtctga aggaagaagg tctagaaaag attggaacgt gggatccagc cagtggcctg 1140
aatatgacag aaagtcaaaa gggaaagcca gcgaacatca cagattcctt atccaatcgt 1200
tctttgattg ttaccaccat tttggaagag ccttatgtcc tttttaagaa gtctgacaaa 1260
cctctctatg gtaatgatcg atttgaaggc tattgcattg atctcctcag agagttatct 1320
acaatccttg gctttacata tgaaattaga cttgtggaag atgggaaata tggagcccag 1380
gatgatgcca atggacaatg gaatggaatg gttcgtgaac taattgatca taaagctgac 1440
cttgcagttg ctccactggc tattacctat gttcgagaga aggtcatcga cttttccaag 1500
ccctttatga cacttggaat aagtattttg taccgcaagc ccaatggtac aaacccaggc 1560
gtcttctcct tcctgaatcc tctctcccct gatatctgga tgtatattct gctggcttac 1620
ttgggtgtca gttgtgtgct ctttgtcata gccaggttta gtccttatga gtggtataat 1680
ccacaccctt gcaaccctga ctcagacgtg gtggaaaaca attttacctt gctaaatagt 1740
ttctggtttg gagttggagc tctcatgcag caaggttctg agctcatgcc caaagcactg 1800
tccaccagga tagtgggagg catttggtgg tttttcacac ttatcatcat ttcttcgtat 1860
actgctaact tagccgcctt tctgacagtg gaacgcatgg aatcccctat tgactctgct 1920
gatgatttag ctaaacaaac caagatagaa tatggagcag tagaggatgg tgcaaccatg 1980
acttttttca agaaatcaaa aatctccacg tatgacaaaa tgtgggcctt tatgagtagc 2040
agaaggcagt cagtgctggt caaaagtaat gaagaaggaa tccagcgagt cctcacctct 2100
gattatgctt tcctaatgga gtcaacaacc atcgagtttg ttacccagcg gaactgtaac 2160
ctgacacaga ttggcggcct tatagactct aaaggttatg gcgttggcac tcccatgggt 2220
tctccatatc gagacaaaat taccatagca attcttcagc tgcaagagga aggcaaactg 2280
catatgatga aggagaaatg gtggaggggc aatggttgcc cagaagagga gagcaaagag 2340
gccagtgccc tgggggttca gaatattggt ggcatcttca ttgttctggc agccggcttg 2400
gtgctttcag tttttgtggc agtgggagaa tttttataca aatccaaaaa aaacgctcaa 2460
ttggaaaaga ggtccttctg tagtgccatg gtagaagaat tgaggatgtc cctgaagtgc 2520
cagcgtcggt taaaacataa gccacaggcc ccagttattg tgaaaacaga agaagttatc 2580
aacatgcaca catttaacga cagaaggttg ccaggtaaag aaaccatggc a 2631
<210> 126
<211> 293
<212> DNA
<213> Homo sapiens
<400> 126
gctcgcgcgg ccggacattg tgggtgtgcg tgctggattt ctcccggatg ctctccgact 60
aacatggatg tcccaccatt ccttgcagtg gaaggttgtt ccttggcgca gtgagtgaag 120
aacatgcagc gattgctaat gggtttggga agcggagact ccttcctctc tctatgacca 180
tgccgtgatc gtgtctgcgg tcaccactcg acgcatcctc atttctaccc gaacccagga 240
gccgaacgct agatcgggga agtgggtgcc gtgcgtgtgg gcacagaaac acc 293
<210> 127
<211> 1572
<212> DNA
<213> Homo sapiens
<400> 127
agctgggagg ccaaacaccc aagcacaaac tgtcgtcttt ttccaaacaa tttagccaga 60
atgtttcctg tggaaatatg caacctgtgc aaaataaaat gagttacctc atgccgctgt 120
gtctatgaac tagagactct gtgatctaag cagttgcaat gatcagactt gatttacaag 180
catcatggat caaccaagtt acacggggtt acactgttaa tcatgggttc ctcccttctt 240
ctgagtgaat gttaacatgc gcattttgtg gctgatttca aatgcagtcc agtgagaaat 300
tacaggttcc ttttgaagct caactgttgc caggagatgg aatatcaatg cccaacaggg 360
caaccaataa aagtgtcact aagaatataa atatttggaa tcagcaaaaa ctgtagtgtt 420
acaggaaaca gtacagtctt ctgaacaccc agatcataga ggtgatgatg ttactagccc 480
ccaactactc agtataatta ttgtctgaat gcaaagtatg tgtttatagg atgtgaaaaa 540
atgtaatgca aaacaaattt gaatcccatg gcagttggaa tataaagcag atgttcatca 600
cttattttcc ttttttcttt tcttattttt ttttttgaca gtctgtgtca ctgattgaga 660
tagaaatgcc aattatcaag gaaataatgt tttcttaagt tccctaaggc agaagattta 720
acatgcaatt ctaccagatc ccttcctatt cccccaacac cttttctcta acccccatat 780
cccaaataat aataataata ataataataa taataataat aataataaaa gcagttggtt 840
cagtgattct gaattaaaag gataatgttt tgcaatgttc aagttgtaaa aactggccga 900
gtattggctg tgtggaagac taaagctttc attctaacat tcagacatag caatccaaac 960
ccttgttcct gctgtaaatg aacttgatgg agcatgggca gatttcagtg atacgagaaa 1020
ggggactggt catctataga aaaatctgtg agagaacttg gaagtggact gcgtttatca 1080
atacagtcac aatgttaaat gaacaaaatt cttgaacagt tttttttcaa aaaatgttca 1140
ggtttatttg tggaaatgca agatttctat gaaaatagtt tttgtatgga aatttttgta 1200
atacttttta tcaacaaaac aagaacatgt gttcctgtca ggggtgtgat gtcaagcatg 1260
aatggtagtg cgtgtgcacc accaacgttt ggtgaaaact atttttatca agaaaaaagg 1320
aatcatagaa gagaaatatt ttcaagttag ataatataaa agctaggtgc actaccacca 1380
ctgcttacca tgccacaccc ctggtttcca cgaggctgac aacatactgt aatgaacaat 1440
tgtgtgtaaa atggtaaaag acacagacct cttgacaaca ttgtgataac agttgagtgc 1500
acacagtttg ctgtttgaat ccaatgcaca aaattaaaaa aaatcattaa aactatgttc 1560
attttacttt ca 1572
<210> 128
<211> 93
<212> DNA
<213> Homo sapiens
<400> 128
atgaagatta ttttcccgat tctaagtaat ccagtcttca ggcgcaccgt taaactcctg 60
ctctgtttac tgtggattgg atattctcaa gga 93
<210> 129
<211> 408
<212> DNA
<213> Homo sapiens
<400> 129
gctcgcgcgg ccggacattg tgggtgtgcg tgctggattt ctcccggatg ctctccgact 60
aacatggatg tcccaccatt ccttgcagtg gaaggttgtt ccttggcgca gtgagtgaag 120
aacatgcagc gattgctaat gggtttggga agcggagact ccttcctctc tctatgacca 180
tgccgtgatc gtgtctgcgg tcaccactcg acgcatcctc atttctaccc gaacccagga 240
gccgaacgct agatcgggga agtgggtgcc gtgcgtgtgg gcacagaaac accatgaaga 300
ttattttccc gattctaagt aatccagtct tcaggcgcac cgttaaactc ctgctctgtt 360
tactgtggat tggatattct caaggaacca cacatgtatt aagatttg 408
<210> 130
<211> 168
<212> DNA
<213> Homo sapiens
<400> 130
gtggtatttt tgaatatgtg gaatctggcc caatgggagc tgaggaactt gcattcagat 60
ttgctgtgaa cacaattaac agaaacagaa cattgctacc caatactacc cttacctatg 120
atacccagaa gataaacctt tatgatagtt ttgaagcatc caagaaag 168
<210> 131
<211> 258
<212> DNA
<213> Homo sapiens
<400> 131
cctgtgatca gctgtctctt ggggtggctg ccatcttcgg gccttcacac agctcatcag 60
caaacgcagt gcagtccatc tgcaatgctc tgggagttcc ccacatacag acccgctgga 120
agcaccaggt gtcagacaac aaagattcct tctatgtcag tctctaccca gacttctctt 180
cactcagccg tgccatttta gacctggtgc agtttttcaa gtggaaaacc gtcacggttg 240
tgtatgatga cagcactg 258
<210> 132
<211> 182
<212> DNA
<213> Homo sapiens
<400> 132
gtctcattcg tttgcaagag ctcatcaaag ctccatcaag gtataatctt cgactcaaaa 60
ttcgtcagtt acctgctgat acaaaggatg caaaaccctt actaaaagaa atgaaaagag 120
gcaaggagtt tcatgtaatc tttgattgta gccatgaaat ggcagcaggc attttaaaac 180
ag 182
<210> 133
<211> 54
<212> DNA
<213> Homo sapiens
<400> 133
gcattagcta tgggaatgat gacagaatac tatcattata tctttaccac tctg 54
<210> 134
<211> 174
<212> DNA
<213> Homo sapiens
<400> 134
gacctctttg ctcttgatgt tgagccctac cgatacagtg gtgttaacat gacagggttc 60
agaatattaa atacagaaaa tacccaagtc tcctccatca ttgaaaagtg gtcgatggaa 120
cgattgcagg cacctccgaa acccgattca ggtttgctgg atggatttat gacg 174
<210> 135
<211> 144
<212> DNA
<213> Homo sapiens
<400> 135
actgatgctg ctctaatgta tgatgctgtg catgtggtgt ctgtggccgt tcaacagttt 60
ccccagatga cagtcagttc cttgcagtgt aatcgacata aaccctggcg cttcgggacc 120
cgctttatga gtctaattaa agag 144
<210> 136
<211> 108
<212> DNA
<213> Homo sapiens
<400> 136
gcacattggg aaggcctcac aggcagaata actttcaaca aaaccaatgg cttgagaaca 60
gattttgatt tggatgtgat cagtctgaag gaagaaggtc tagaaaag 108
<210> 137
<211> 114
<212> DNA
<213> Homo sapiens
<400> 137
attggaacgt gggatccagc cagtggcctg aatatgacag aaagtcaaaa gggaaagcca 60
gcgaacatca cagattcctt atccaatcgt tctttgattg ttaccaccat tttg 114
<210> 138
<211> 207
<212> DNA
<213> Homo sapiens
<400> 138
gaagagcctt atgtcctttt taagaagtct gacaaacctc tctatggtaa tgatcgattt 60
gaaggctatt gcattgatct cctcagagag ttatctacaa tccttggctt tacatatgaa 120
attagacttg tggaagatgg gaaatatgga gcccaggatg atgccaatgg acaatggaat 180
ggaatggttc gtgaactaat tgatcat 207
<210> 139
<211> 224
<212> DNA
<213> Homo sapiens
<400> 139
aaagctgacc ttgcagttgc tccactggct attacctatg ttcgagagaa ggtcatcgac 60
ttttccaagc cctttatgac acttggaata agtattttgt accgcaagcc caatggtaca 120
aacccaggcg tcttctcctt cctgaatcct ctctcccctg atatctggat gtatattctg 180
ctggcttact tgggtgtcag ttgtgtgctc tttgtcatag ccag 224
<210> 140
<211> 119
<212> DNA
<213> Homo sapiens
<400> 140
gtttagtcct tatgagtggt ataatccaca cccttgcaac cctgactcag acgtggtgga 60
aaacaatttt accttgctaa atagtttctg gtttggagtt ggagctctca tgcagcaag 119
<210> 141
<211> 218
<212> DNA
<213> Homo sapiens
<400> 141
gttctgagct catgcccaaa gcactgtcca ccaggatagt gggaggcatt tggtggtttt 60
tcacacttat catcatttct tcgtatactg ctaacttagc cgcctttctg acagtggaac 120
gcatggaatc ccctattgac tctgctgatg atttagctaa acaaaccaag atagaatatg 180
gagcagtaga ggatggtgca accatgactt ttttcaag 218
<210> 142
<211> 226
<212> DNA
<213> Homo sapiens
<400> 142
aaatcaaaaa tctccacgta tgacaaaatg tgggccttta tgagtagcag aaggcagtca 60
gtgctggtca aaagtaatga agaaggaatc cagcgagtcc tcacctctga ttatgctttc 120
ctaatggagt caacaaccat cgagtttgtt acccagcgga actgtaacct gacacagatt 180
ggcggcctta tagactctaa aggttatggc gttggcactc ccatgg 226
<210> 143
<211> 251
<212> DNA
<213> Homo sapiens
<400> 143
gttctccata tcgagacaaa attaccatag caattcttca gctgcaagag gaaggcaaac 60
tgcatatgat gaaggagaaa tggtggaggg gcaatggttg cccagaagag gagagcaaag 120
aggccagtgc cctgggggtt cagaatattg gtggcatctt cattgttctg gcagccggct 180
tggtgctttc agtttttgtg gcagtgggag aatttttata caaatccaaa aaaaacgctc 240
aattggaaaa g 251
<210> 144
<211> 1737
<212> DNA
<213> Homo sapiens
<400> 144
aggtccttct gtagtgccat ggtagaagaa ttgaggatgt ccctgaagtg ccagcgtcgg 60
ttaaaacata agccacaggc cccagttatt gtgaaaacag aagaagttat caacatgcac 120
acatttaacg acagaaggtt gccaggtaaa gaaaccatgg cataaagctg ggaggccaaa 180
cacccaagca caaactgtcg tctttttcca aacaatttag ccagaatgtt tcctgtggaa 240
atatgcaacc tgtgcaaaat aaaatgagtt acctcatgcc gctgtgtcta tgaactagag 300
actctgtgat ctaagcagtt gcaatgatca gacttgattt acaagcatca tggatcaacc 360
aagttacacg gggttacact gttaatcatg ggttcctccc ttcttctgag tgaatgttaa 420
catgcgcatt ttgtggctga tttcaaatgc agtccagtga gaaattacag gttccttttg 480
aagctcaact gttgccagga gatggaatat caatgcccaa cagggcaacc aataaaagtg 540
tcactaagaa tataaatatt tggaatcagc aaaaactgta gtgttacagg aaacagtaca 600
gtcttctgaa cacccagatc atagaggtga tgatgttact agcccccaac tactcagtat 660
aattattgtc tgaatgcaaa gtatgtgttt ataggatgtg aaaaaatgta atgcaaaaca 720
aatttgaatc ccatggcagt tggaatataa agcagatgtt catcacttat tttccttttt 780
tcttttctta tttttttttt tgacagtctg tgtcactgat tgagatagaa atgccaatta 840
tcaaggaaat aatgttttct taagttccct aaggcagaag atttaacatg caattctacc 900
agatcccttc ctattccccc aacacctttt ctctaacccc catatcccaa ataataataa 960
taataataat aataataata ataataataa taaaagcagt tggttcagtg attctgaatt 1020
aaaaggataa tgttttgcaa tgttcaagtt gtaaaaactg gccgagtatt ggctgtgtgg 1080
aagactaaag ctttcattct aacattcaga catagcaatc caaacccttg ttcctgctgt 1140
aaatgaactt gatggagcat gggcagattt cagtgatacg agaaagggga ctggtcatct 1200
atagaaaaat ctgtgagaga acttggaagt ggactgcgtt tatcaataca gtcacaatgt 1260
taaatgaaca aaattcttga acagtttttt ttcaaaaaat gttcaggttt atttgtggaa 1320
atgcaagatt tctatgaaaa tagtttttgt atggaaattt ttgtaatact ttttatcaac 1380
aaaacaagaa catgtgttcc tgtcaggggt gtgatgtcaa gcatgaatgg tagtgcgtgt 1440
gcaccaccaa cgtttggtga aaactatttt tatcaagaaa aaaggaatca tagaagagaa 1500
atattttcaa gttagataat ataaaagcta ggtgcactac caccactgct taccatgcca 1560
cacccctggt ttccacgagg ctgacaacat actgtaatga acaattgtgt gtaaaatggt 1620
aaaagacaca gacctcttga caacattgtg ataacagttg agtgcacaca gtttgctgtt 1680
tgaatccaat gcacaaaatt aaaaaaaatc attaaaacta tgttcatttt actttca 1737
<210> 145
<211> 31
<212> DNA
<213> Homo sapiens
<400> 145
cagaacattg ctacccaata ctacccttac c 31
<210> 146
<211> 27
<212> DNA
<213> Homo sapiens
<400> 146
taccgataca gtggtgttaa catgaca 27
<210> 147
<211> 41
<212> DNA
<213> Homo sapiens
<400> 147
caggcacctc cgaaacccga ttcaggtttg ctggatggat t 41
<210> 148
<211> 27
<212> DNA
<213> Homo sapiens
<400> 148
aaaagggaaa gccagcgaac atcacag 27
<210> 149
<211> 65
<212> DNA
<213> Homo sapiens
<400> 149
atttgaaggc tattgcattg atctcctcag agagttatct acaatccttg gctttacata 60
tgaaa 65
<210> 150
<211> 64
<212> DNA
<213> Homo sapiens
<400> 150
gatgatgcca atggacaatg gaatggaatg gttcgtgaac taattgatca taaagctgac 60
cttg 64
<210> 151
<211> 24
<212> DNA
<213> Homo sapiens
<400> 151
gggcaatggt tgcccagaag agga 24
<210> 152
<211> 40
<212> DNA
<213> Homo sapiens
<400> 152
ttaaaacata agccacaggc cccagttatt gtgaaaacag 40
<210> 153
<211> 27
<212> DNA
<213> Homo sapiens
<400> 153
aaacaattta gccagaatgt ttcctgt 27
<210> 154
<211> 23
<212> DNA
<213> Homo sapiens
<400> 154
gtctatgaac tagagactct gtg 23
<210> 155
<211> 32
<212> DNA
<213> Homo sapiens
<400> 155
aaccaataaa agtgtcacta agaatataaa ta 32
<210> 156
<211> 69
<212> DNA
<213> Homo sapiens
<400> 156
ctaaccccca tatcccaaat aataataata ataataataa taataataat aataataata 60
aaagcagtt 69
<210> 157
<211> 43
<212> DNA
<213> Homo sapiens
<400> 157
aaaattaaaa aaaatcatta aaactatgtt cattttactt tca 43
<210> 158
<211> 24
<212> DNA
<213> Homo sapiens
<400> 158
caagttagat aatataaaag ctag 24
<210> 159
<211> 79
<212> DNA
<213> Homo sapiens
<400> 159
tttggtggtt tttcacactt atcatcattt cttcgtatac tgctaactta gccgcctttc 60
tgacagtgga acgcatgga 79
<210> 160
<211> 37
<212> DNA
<213> Homo sapiens
<400> 160
ggtgcaacca tgactttttt caagaaatca aaaatct 37
<210> 161
<211> 45
<212> DNA
<213> Homo sapiens
<400> 161
aggttccttt tgaagctcaa ctgttgccag gagatggaat atcaa 45
<210> 162
<211> 53
<212> DNA
<213> Homo sapiens
<400> 162
catggcagtt ggaatataaa gcagatgttc atcacttatt ttcctttttt ctt 53
<210> 163
<211> 28
<212> DNA
<213> Homo sapiens
<400> 163
atgccaatta tcaaggaaat aatgtttt 28
<210> 164
<211> 20
<212> DNA
<213> Homo sapiens
<400> 164
cagaacattg ctacccaata 20
<210> 165
<211> 20
<212> DNA
<213> Homo sapiens
<400> 165
agaacattgc tacccaatac 20
<210> 166
<211> 20
<212> DNA
<213> Homo sapiens
<400> 166
gaacattgct acccaatact 20
<210> 167
<211> 20
<212> DNA
<213> Homo sapiens
<400> 167
aacattgcta cccaatacta 20
<210> 168
<211> 20
<212> DNA
<213> Homo sapiens
<400> 168
acattgctac ccaatactac 20
<210> 169
<211> 20
<212> DNA
<213> Homo sapiens
<400> 169
cattgctacc caatactacc 20
<210> 170
<211> 20
<212> DNA
<213> Homo sapiens
<400> 170
attgctaccc aatactaccc 20
<210> 171
<211> 20
<212> DNA
<213> Homo sapiens
<400> 171
ttgctaccca atactaccct 20
<210> 172
<211> 20
<212> DNA
<213> Homo sapiens
<400> 172
tgctacccaa tactaccctt 20
<210> 173
<211> 20
<212> DNA
<213> Homo sapiens
<400> 173
gctacccaat actaccctta 20
<210> 174
<211> 20
<212> DNA
<213> Homo sapiens
<400> 174
ctacccaata ctacccttac 20
<210> 175
<211> 20
<212> DNA
<213> Homo sapiens
<400> 175
tacccaatac tacccttacc 20
<210> 176
<211> 20
<212> DNA
<213> Homo sapiens
<400> 176
taccgataca gtggtgttaa 20
<210> 177
<211> 20
<212> DNA
<213> Homo sapiens
<400> 177
accgatacag tggtgttaac 20
<210> 178
<211> 20
<212> DNA
<213> Homo sapiens
<400> 178
ccgatacagt ggtgttaaca 20
<210> 179
<211> 20
<212> DNA
<213> Homo sapiens
<400> 179
cgatacagtg gtgttaacat 20
<210> 180
<211> 20
<212> DNA
<213> Homo sapiens
<400> 180
gatacagtgg tgttaacatg 20
<210> 181
<211> 20
<212> DNA
<213> Homo sapiens
<400> 181
atacagtggt gttaacatga 20
<210> 182
<211> 20
<212> DNA
<213> Homo sapiens
<400> 182
tacagtggtg ttaacatgac 20
<210> 183
<211> 20
<212> DNA
<213> Homo sapiens
<400> 183
acagtggtgt taacatgaca 20
<210> 184
<211> 20
<212> DNA
<213> Homo sapiens
<400> 184
caggcacctc cgaaacccga 20
<210> 185
<211> 20
<212> DNA
<213> Homo sapiens
<400> 185
aggcacctcc gaaacccgat 20
<210> 186
<211> 20
<212> DNA
<213> Homo sapiens
<400> 186
ggcacctccg aaacccgatt 20
<210> 187
<211> 20
<212> DNA
<213> Homo sapiens
<400> 187
gcacctccga aacccgattc 20
<210> 188
<211> 20
<212> DNA
<213> Homo sapiens
<400> 188
cacctccgaa acccgattca 20
<210> 189
<211> 20
<212> DNA
<213> Homo sapiens
<400> 189
acctccgaaa cccgattcag 20
<210> 190
<211> 20
<212> DNA
<213> Homo sapiens
<400> 190
cctccgaaac ccgattcagg 20
<210> 191
<211> 20
<212> DNA
<213> Homo sapiens
<400> 191
ctccgaaacc cgattcaggt 20
<210> 192
<211> 20
<212> DNA
<213> Homo sapiens
<400> 192
tccgaaaccc gattcaggtt 20
<210> 193
<211> 20
<212> DNA
<213> Homo sapiens
<400> 193
ccgaaacccg attcaggttt 20
<210> 194
<211> 20
<212> DNA
<213> Homo sapiens
<400> 194
cgaaacccga ttcaggtttg 20
<210> 195
<211> 20
<212> DNA
<213> Homo sapiens
<400> 195
gaaacccgat tcaggtttgc 20
<210> 196
<211> 20
<212> DNA
<213> Homo sapiens
<400> 196
aaacccgatt caggtttgct 20
<210> 197
<211> 20
<212> DNA
<213> Homo sapiens
<400> 197
aacccgattc aggtttgctg 20
<210> 198
<211> 20
<212> DNA
<213> Homo sapiens
<400> 198
acccgattca ggtttgctgg 20
<210> 199
<211> 20
<212> DNA
<213> Homo sapiens
<400> 199
cccgattcag gtttgctgga 20
<210> 200
<211> 20
<212> DNA
<213> Homo sapiens
<400> 200
ccgattcagg tttgctggat 20
<210> 201
<211> 20
<212> DNA
<213> Homo sapiens
<400> 201
cgattcaggt ttgctggatg 20
<210> 202
<211> 20
<212> DNA
<213> Homo sapiens
<400> 202
gattcaggtt tgctggatgg 20
<210> 203
<211> 20
<212> DNA
<213> Homo sapiens
<400> 203
attcaggttt gctggatgga 20
<210> 204
<211> 20
<212> DNA
<213> Homo sapiens
<400> 204
ttcaggtttg ctggatggat 20
<210> 205
<211> 20
<212> DNA
<213> Homo sapiens
<400> 205
tcaggtttgc tggatggatt 20
<210> 206
<211> 20
<212> DNA
<213> Homo sapiens
<400> 206
aaaagggaaa gccagcgaac 20
<210> 207
<211> 20
<212> DNA
<213> Homo sapiens
<400> 207
aaagggaaag ccagcgaaca 20
<210> 208
<211> 20
<212> DNA
<213> Homo sapiens
<400> 208
aagggaaagc cagcgaacat 20
<210> 209
<211> 20
<212> DNA
<213> Homo sapiens
<400> 209
agggaaagcc agcgaacatc 20
<210> 210
<211> 20
<212> DNA
<213> Homo sapiens
<400> 210
gggaaagcca gcgaacatca 20
<210> 211
<211> 20
<212> DNA
<213> Homo sapiens
<400> 211
ggaaagccag cgaacatcac 20
<210> 212
<211> 20
<212> DNA
<213> Homo sapiens
<400> 212
gaaagccagc gaacatcaca 20
<210> 213
<211> 20
<212> DNA
<213> Homo sapiens
<400> 213
aaagccagcg aacatcacag 20
<210> 214
<211> 20
<212> DNA
<213> Homo sapiens
<400> 214
atttgaaggc tattgcattg 20
<210> 215
<211> 20
<212> DNA
<213> Homo sapiens
<400> 215
tttgaaggct attgcattga 20
<210> 216
<211> 20
<212> DNA
<213> Homo sapiens
<400> 216
ttgaaggcta ttgcattgat 20
<210> 217
<211> 20
<212> DNA
<213> Homo sapiens
<400> 217
tgaaggctat tgcattgatc 20
<210> 218
<211> 20
<212> DNA
<213> Homo sapiens
<400> 218
gaaggctatt gcattgatct 20
<210> 219
<211> 20
<212> DNA
<213> Homo sapiens
<400> 219
aaggctattg cattgatctc 20
<210> 220
<211> 20
<212> DNA
<213> Homo sapiens
<400> 220
aggctattgc attgatctcc 20
<210> 221
<211> 20
<212> DNA
<213> Homo sapiens
<400> 221
ggctattgca ttgatctcct 20
<210> 222
<211> 20
<212> DNA
<213> Homo sapiens
<400> 222
gctattgcat tgatctcctc 20
<210> 223
<211> 20
<212> DNA
<213> Homo sapiens
<400> 223
ctattgcatt gatctcctca 20
<210> 224
<211> 20
<212> DNA
<213> Homo sapiens
<400> 224
tattgcattg atctcctcag 20
<210> 225
<211> 20
<212> DNA
<213> Homo sapiens
<400> 225
attgcattga tctcctcaga 20
<210> 226
<211> 20
<212> DNA
<213> Homo sapiens
<400> 226
ttgcattgat ctcctcagag 20
<210> 227
<211> 20
<212> DNA
<213> Homo sapiens
<400> 227
tgcattgatc tcctcagaga 20
<210> 228
<211> 20
<212> DNA
<213> Homo sapiens
<400> 228
gcattgatct cctcagagag 20
<210> 229
<211> 20
<212> DNA
<213> Homo sapiens
<400> 229
cattgatctc ctcagagagt 20
<210> 230
<211> 20
<212> DNA
<213> Homo sapiens
<400> 230
attgatctcc tcagagagtt 20
<210> 231
<211> 20
<212> DNA
<213> Homo sapiens
<400> 231
ttgatctcct cagagagtta 20
<210> 232
<211> 20
<212> DNA
<213> Homo sapiens
<400> 232
tgatctcctc agagagttat 20
<210> 233
<211> 20
<212> DNA
<213> Homo sapiens
<400> 233
gatctcctca gagagttatc 20
<210> 234
<211> 20
<212> DNA
<213> Homo sapiens
<400> 234
atctcctcag agagttatct 20
<210> 235
<211> 20
<212> DNA
<213> Homo sapiens
<400> 235
tctcctcaga gagttatcta 20
<210> 236
<211> 20
<212> DNA
<213> Homo sapiens
<400> 236
ctcctcagag agttatctac 20
<210> 237
<211> 20
<212> DNA
<213> Homo sapiens
<400> 237
tcctcagaga gttatctaca 20
<210> 238
<211> 20
<212> DNA
<213> Homo sapiens
<400> 238
cctcagagag ttatctacaa 20
<210> 239
<211> 20
<212> DNA
<213> Homo sapiens
<400> 239
ctcagagagt tatctacaat 20
<210> 240
<211> 20
<212> DNA
<213> Homo sapiens
<400> 240
tcagagagtt atctacaatc 20
<210> 241
<211> 20
<212> DNA
<213> Homo sapiens
<400> 241
cagagagtta tctacaatcc 20
<210> 242
<211> 20
<212> DNA
<213> Homo sapiens
<400> 242
agagagttat ctacaatcct 20
<210> 243
<211> 20
<212> DNA
<213> Homo sapiens
<400> 243
gagagttatc tacaatcctt 20
<210> 244
<211> 20
<212> DNA
<213> Homo sapiens
<400> 244
agagttatct acaatccttg 20
<210> 245
<211> 20
<212> DNA
<213> Homo sapiens
<400> 245
gagttatcta caatccttgg 20
<210> 246
<211> 20
<212> DNA
<213> Homo sapiens
<400> 246
agttatctac aatccttggc 20
<210> 247
<211> 20
<212> DNA
<213> Homo sapiens
<400> 247
gttatctaca atccttggct 20
<210> 248
<211> 20
<212> DNA
<213> Homo sapiens
<400> 248
ttatctacaa tccttggctt 20
<210> 249
<211> 20
<212> DNA
<213> Homo sapiens
<400> 249
tatctacaat ccttggcttt 20
<210> 250
<211> 20
<212> DNA
<213> Homo sapiens
<400> 250
atctacaatc cttggcttta 20
<210> 251
<211> 20
<212> DNA
<213> Homo sapiens
<400> 251
tctacaatcc ttggctttac 20
<210> 252
<211> 20
<212> DNA
<213> Homo sapiens
<400> 252
ctacaatcct tggctttaca 20
<210> 253
<211> 20
<212> DNA
<213> Homo sapiens
<400> 253
tacaatcctt ggctttacat 20
<210> 254
<211> 20
<212> DNA
<213> Homo sapiens
<400> 254
acaatccttg gctttacata 20
<210> 255
<211> 20
<212> DNA
<213> Homo sapiens
<400> 255
caatccttgg ctttacatat 20
<210> 256
<211> 20
<212> DNA
<213> Homo sapiens
<400> 256
aatccttggc tttacatatg 20
<210> 257
<211> 20
<212> DNA
<213> Homo sapiens
<400> 257
atccttggct ttacatatga 20
<210> 258
<211> 20
<212> DNA
<213> Homo sapiens
<400> 258
tccttggctt tacatatgaa 20
<210> 259
<211> 20
<212> DNA
<213> Homo sapiens
<400> 259
ccttggcttt acatatgaaa 20
<210> 260
<211> 20
<212> DNA
<213> Homo sapiens
<400> 260
gatgatgcca atggacaatg 20
<210> 261
<211> 20
<212> DNA
<213> Homo sapiens
<400> 261
atgatgccaa tggacaatgg 20
<210> 262
<211> 20
<212> DNA
<213> Homo sapiens
<400> 262
tgatgccaat ggacaatgga 20
<210> 263
<211> 20
<212> DNA
<213> Homo sapiens
<400> 263
gatgccaatg gacaatggaa 20
<210> 264
<211> 20
<212> DNA
<213> Homo sapiens
<400> 264
atgccaatgg acaatggaat 20
<210> 265
<211> 20
<212> DNA
<213> Homo sapiens
<400> 265
tgccaatgga caatggaatg 20
<210> 266
<211> 20
<212> DNA
<213> Homo sapiens
<400> 266
gccaatggac aatggaatgg 20
<210> 267
<211> 20
<212> DNA
<213> Homo sapiens
<400> 267
ccaatggaca atggaatgga 20
<210> 268
<211> 20
<212> DNA
<213> Homo sapiens
<400> 268
caatggacaa tggaatggaa 20
<210> 269
<211> 20
<212> DNA
<213> Homo sapiens
<400> 269
aatggacaat ggaatggaat 20
<210> 270
<211> 20
<212> DNA
<213> Homo sapiens
<400> 270
atggacaatg gaatggaatg 20
<210> 271
<211> 20
<212> DNA
<213> Homo sapiens
<400> 271
tggacaatgg aatggaatgg 20
<210> 272
<211> 20
<212> DNA
<213> Homo sapiens
<400> 272
ggacaatgga atggaatggt 20
<210> 273
<211> 20
<212> DNA
<213> Homo sapiens
<400> 273
gacaatggaa tggaatggtt 20
<210> 274
<211> 20
<212> DNA
<213> Homo sapiens
<400> 274
acaatggaat ggaatggttc 20
<210> 275
<211> 20
<212> DNA
<213> Homo sapiens
<400> 275
caatggaatg gaatggttcg 20
<210> 276
<211> 20
<212> DNA
<213> Homo sapiens
<400> 276
aatggaatgg aatggttcgt 20
<210> 277
<211> 20
<212> DNA
<213> Homo sapiens
<400> 277
atggaatgga atggttcgtg 20
<210> 278
<211> 20
<212> DNA
<213> Homo sapiens
<400> 278
tggaatggaa tggttcgtga 20
<210> 279
<211> 20
<212> DNA
<213> Homo sapiens
<400> 279
ggaatggaat ggttcgtgaa 20
<210> 280
<211> 20
<212> DNA
<213> Homo sapiens
<400> 280
gaatggaatg gttcgtgaac 20
<210> 281
<211> 20
<212> DNA
<213> Homo sapiens
<400> 281
aatggaatgg ttcgtgaact 20
<210> 282
<211> 20
<212> DNA
<213> Homo sapiens
<400> 282
atggaatggt tcgtgaacta 20
<210> 283
<211> 20
<212> DNA
<213> Homo sapiens
<400> 283
tggaatggtt cgtgaactaa 20
<210> 284
<211> 20
<212> DNA
<213> Homo sapiens
<400> 284
ggaatggttc gtgaactaat 20
<210> 285
<211> 20
<212> DNA
<213> Homo sapiens
<400> 285
gaatggttcg tgaactaatt 20
<210> 286
<211> 20
<212> DNA
<213> Homo sapiens
<400> 286
aatggttcgt gaactaattg 20
<210> 287
<211> 20
<212> DNA
<213> Homo sapiens
<400> 287
atggttcgtg aactaattga 20
<210> 288
<211> 20
<212> DNA
<213> Homo sapiens
<400> 288
tggttcgtga actaattgat 20
<210> 289
<211> 20
<212> DNA
<213> Homo sapiens
<400> 289
ggttcgtgaa ctaattgatc 20
<210> 290
<211> 20
<212> DNA
<213> Homo sapiens
<400> 290
gttcgtgaac taattgatca 20
<210> 291
<211> 20
<212> DNA
<213> Homo sapiens
<400> 291
ttcgtgaact aattgatcat 20
<210> 292
<211> 20
<212> DNA
<213> Homo sapiens
<400> 292
tcgtgaacta attgatcata 20
<210> 293
<211> 20
<212> DNA
<213> Homo sapiens
<400> 293
cgtgaactaa ttgatcataa 20
<210> 294
<211> 20
<212> DNA
<213> Homo sapiens
<400> 294
gtgaactaat tgatcataaa 20
<210> 295
<211> 20
<212> DNA
<213> Homo sapiens
<400> 295
tgaactaatt gatcataaag 20
<210> 296
<211> 20
<212> DNA
<213> Homo sapiens
<400> 296
gaactaattg atcataaagc 20
<210> 297
<211> 20
<212> DNA
<213> Homo sapiens
<400> 297
aactaattga tcataaagct 20
<210> 298
<211> 20
<212> DNA
<213> Homo sapiens
<400> 298
actaattgat cataaagctg 20
<210> 299
<211> 20
<212> DNA
<213> Homo sapiens
<400> 299
ctaattgatc ataaagctga 20
<210> 300
<211> 20
<212> DNA
<213> Homo sapiens
<400> 300
taattgatca taaagctgac 20
<210> 301
<211> 20
<212> DNA
<213> Homo sapiens
<400> 301
aattgatcat aaagctgacc 20
<210> 302
<211> 20
<212> DNA
<213> Homo sapiens
<400> 302
attgatcata aagctgacct 20
<210> 303
<211> 20
<212> DNA
<213> Homo sapiens
<400> 303
ttgatcataa agctgacctt 20
<210> 304
<211> 20
<212> DNA
<213> Homo sapiens
<400> 304
tgatcataaa gctgaccttg 20
<210> 305
<211> 20
<212> DNA
<213> Homo sapiens
<400> 305
gggcaatggt tgcccagaag 20
<210> 306
<211> 20
<212> DNA
<213> Homo sapiens
<400> 306
ggcaatggtt gcccagaaga 20
<210> 307
<211> 20
<212> DNA
<213> Homo sapiens
<400> 307
gcaatggttg cccagaagag 20
<210> 308
<211> 20
<212> DNA
<213> Homo sapiens
<400> 308
caatggttgc ccagaagagg 20
<210> 309
<211> 20
<212> DNA
<213> Homo sapiens
<400> 309
aatggttgcc cagaagagga 20
<210> 310
<211> 20
<212> DNA
<213> Homo sapiens
<400> 310
ttaaaacata agccacaggc 20
<210> 311
<211> 20
<212> DNA
<213> Homo sapiens
<400> 311
taaaacataa gccacaggcc 20
<210> 312
<211> 20
<212> DNA
<213> Homo sapiens
<400> 312
aaaacataag ccacaggccc 20
<210> 313
<211> 20
<212> DNA
<213> Homo sapiens
<400> 313
aaacataagc cacaggcccc 20
<210> 314
<211> 20
<212> DNA
<213> Homo sapiens
<400> 314
aacataagcc acaggcccca 20
<210> 315
<211> 20
<212> DNA
<213> Homo sapiens
<400> 315
acataagcca caggccccag 20
<210> 316
<211> 20
<212> DNA
<213> Homo sapiens
<400> 316
cataagccac aggccccagt 20
<210> 317
<211> 20
<212> DNA
<213> Homo sapiens
<400> 317
ataagccaca ggccccagtt 20
<210> 318
<211> 20
<212> DNA
<213> Homo sapiens
<400> 318
taagccacag gccccagtta 20
<210> 319
<211> 20
<212> DNA
<213> Homo sapiens
<400> 319
aagccacagg ccccagttat 20
<210> 320
<211> 20
<212> DNA
<213> Homo sapiens
<400> 320
agccacaggc cccagttatt 20
<210> 321
<211> 20
<212> DNA
<213> Homo sapiens
<400> 321
gccacaggcc ccagttattg 20
<210> 322
<211> 20
<212> DNA
<213> Homo sapiens
<400> 322
ccacaggccc cagttattgt 20
<210> 323
<211> 20
<212> DNA
<213> Homo sapiens
<400> 323
cacaggcccc agttattgtg 20
<210> 324
<211> 20
<212> DNA
<213> Homo sapiens
<400> 324
acaggcccca gttattgtga 20
<210> 325
<211> 20
<212> DNA
<213> Homo sapiens
<400> 325
caggccccag ttattgtgaa 20
<210> 326
<211> 20
<212> DNA
<213> Homo sapiens
<400> 326
aggccccagt tattgtgaaa 20
<210> 327
<211> 20
<212> DNA
<213> Homo sapiens
<400> 327
ggccccagtt attgtgaaaa 20
<210> 328
<211> 20
<212> DNA
<213> Homo sapiens
<400> 328
gccccagtta ttgtgaaaac 20
<210> 329
<211> 20
<212> DNA
<213> Homo sapiens
<400> 329
ccccagttat tgtgaaaaca 20
<210> 330
<211> 20
<212> DNA
<213> Homo sapiens
<400> 330
cccagttatt gtgaaaacag 20
<210> 331
<211> 20
<212> DNA
<213> Homo sapiens
<400> 331
aaacaattta gccagaatgt 20
<210> 332
<211> 20
<212> DNA
<213> Homo sapiens
<400> 332
aacaatttag ccagaatgtt 20
<210> 333
<211> 20
<212> DNA
<213> Homo sapiens
<400> 333
acaatttagc cagaatgttt 20
<210> 334
<211> 20
<212> DNA
<213> Homo sapiens
<400> 334
caatttagcc agaatgtttc 20
<210> 335
<211> 20
<212> DNA
<213> Homo sapiens
<400> 335
aatttagcca gaatgtttcc 20
<210> 336
<211> 20
<212> DNA
<213> Homo sapiens
<400> 336
atttagccag aatgtttcct 20
<210> 337
<211> 20
<212> DNA
<213> Homo sapiens
<400> 337
tttagccaga atgtttcctg 20
<210> 338
<211> 20
<212> DNA
<213> Homo sapiens
<400> 338
ttagccagaa tgtttcctgt 20
<210> 339
<211> 20
<212> DNA
<213> Homo sapiens
<400> 339
gtctatgaac tagagactct 20
<210> 340
<211> 20
<212> DNA
<213> Homo sapiens
<400> 340
tctatgaact agagactctg 20
<210> 341
<211> 20
<212> DNA
<213> Homo sapiens
<400> 341
ctatgaacta gagactctgt 20
<210> 342
<211> 20
<212> DNA
<213> Homo sapiens
<400> 342
tatgaactag agactctgtg 20
<210> 343
<211> 20
<212> DNA
<213> Homo sapiens
<400> 343
aaccaataaa agtgtcacta 20
<210> 344
<211> 20
<212> DNA
<213> Homo sapiens
<400> 344
accaataaaa gtgtcactaa 20
<210> 345
<211> 20
<212> DNA
<213> Homo sapiens
<400> 345
ccaataaaag tgtcactaag 20
<210> 346
<211> 20
<212> DNA
<213> Homo sapiens
<400> 346
caataaaagt gtcactaaga 20
<210> 347
<211> 20
<212> DNA
<213> Homo sapiens
<400> 347
aataaaagtg tcactaagaa 20
<210> 348
<211> 20
<212> DNA
<213> Homo sapiens
<400> 348
ataaaagtgt cactaagaat 20
<210> 349
<211> 20
<212> DNA
<213> Homo sapiens
<400> 349
taaaagtgtc actaagaata 20
<210> 350
<211> 20
<212> DNA
<213> Homo sapiens
<400> 350
aaaagtgtca ctaagaatat 20
<210> 351
<211> 20
<212> DNA
<213> Homo sapiens
<400> 351
aaagtgtcac taagaatata 20
<210> 352
<211> 20
<212> DNA
<213> Homo sapiens
<400> 352
aagtgtcact aagaatataa 20
<210> 353
<211> 20
<212> DNA
<213> Homo sapiens
<400> 353
agtgtcacta agaatataaa 20
<210> 354
<211> 20
<212> DNA
<213> Homo sapiens
<400> 354
gtgtcactaa gaatataaat 20
<210> 355
<211> 20
<212> DNA
<213> Homo sapiens
<400> 355
tgtcactaag aatataaata 20
<210> 356
<211> 20
<212> DNA
<213> Homo sapiens
<400> 356
ctaaccccca tatcccaaat 20
<210> 357
<211> 20
<212> DNA
<213> Homo sapiens
<400> 357
taacccccat atcccaaata 20
<210> 358
<211> 20
<212> DNA
<213> Homo sapiens
<400> 358
aacccccata tcccaaataa 20
<210> 359
<211> 20
<212> DNA
<213> Homo sapiens
<400> 359
acccccatat cccaaataat 20
<210> 360
<211> 20
<212> DNA
<213> Homo sapiens
<400> 360
cccccatatc ccaaataata 20
<210> 361
<211> 20
<212> DNA
<213> Homo sapiens
<400> 361
ccccatatcc caaataataa 20
<210> 362
<211> 20
<212> DNA
<213> Homo sapiens
<400> 362
cccatatccc aaataataat 20
<210> 363
<211> 20
<212> DNA
<213> Homo sapiens
<400> 363
ccatatccca aataataata 20
<210> 364
<211> 20
<212> DNA
<213> Homo sapiens
<400> 364
catatcccaa ataataataa 20
<210> 365
<211> 20
<212> DNA
<213> Homo sapiens
<400> 365
atatcccaaa taataataat 20
<210> 366
<211> 20
<212> DNA
<213> Homo sapiens
<400> 366
tatcccaaat aataataata 20
<210> 367
<211> 20
<212> DNA
<213> Homo sapiens
<400> 367
atcccaaata ataataataa 20
<210> 368
<211> 20
<212> DNA
<213> Homo sapiens
<400> 368
tcccaaataa taataataat 20
<210> 369
<211> 20
<212> DNA
<213> Homo sapiens
<400> 369
cccaaataat aataataata 20
<210> 370
<211> 20
<212> DNA
<213> Homo sapiens
<400> 370
ccaaataata ataataataa 20
<210> 371
<211> 20
<212> DNA
<213> Homo sapiens
<400> 371
caaataataa taataataat 20
<210> 372
<211> 20
<212> DNA
<213> Homo sapiens
<400> 372
aaataataat aataataata 20
<210> 373
<211> 20
<212> DNA
<213> Homo sapiens
<400> 373
aataataata ataataataa 20
<210> 374
<211> 20
<212> DNA
<213> Homo sapiens
<400> 374
ataataataa taataataat 20
<210> 375
<211> 20
<212> DNA
<213> Homo sapiens
<400> 375
taataataat aataataata 20
<210> 376
<211> 20
<212> DNA
<213> Homo sapiens
<400> 376
aataataata ataataataa 20
<210> 377
<211> 20
<212> DNA
<213> Homo sapiens
<400> 377
ataataataa taataataat 20
<210> 378
<211> 20
<212> DNA
<213> Homo sapiens
<400> 378
taataataat aataataata 20
<210> 379
<211> 20
<212> DNA
<213> Homo sapiens
<400> 379
aataataata ataataataa 20
<210> 380
<211> 20
<212> DNA
<213> Homo sapiens
<400> 380
ataataataa taataataat 20
<210> 381
<211> 20
<212> DNA
<213> Homo sapiens
<400> 381
taataataat aataataata 20
<210> 382
<211> 20
<212> DNA
<213> Homo sapiens
<400> 382
aataataata ataataataa 20
<210> 383
<211> 20
<212> DNA
<213> Homo sapiens
<400> 383
ataataataa taataataat 20
<210> 384
<211> 20
<212> DNA
<213> Homo sapiens
<400> 384
taataataat aataataata 20
<210> 385
<211> 20
<212> DNA
<213> Homo sapiens
<400> 385
aataataata ataataataa 20
<210> 386
<211> 20
<212> DNA
<213> Homo sapiens
<400> 386
ataataataa taataataat 20
<210> 387
<211> 20
<212> DNA
<213> Homo sapiens
<400> 387
taataataat aataataata 20
<210> 388
<211> 20
<212> DNA
<213> Homo sapiens
<400> 388
aataataata ataataataa 20
<210> 389
<211> 20
<212> DNA
<213> Homo sapiens
<400> 389
ataataataa taataataat 20
<210> 390
<211> 20
<212> DNA
<213> Homo sapiens
<400> 390
taataataat aataataata 20
<210> 391
<211> 20
<212> DNA
<213> Homo sapiens
<400> 391
aataataata ataataataa 20
<210> 392
<211> 20
<212> DNA
<213> Homo sapiens
<400> 392
ataataataa taataataat 20
<210> 393
<211> 20
<212> DNA
<213> Homo sapiens
<400> 393
taataataat aataataata 20
<210> 394
<211> 20
<212> DNA
<213> Homo sapiens
<400> 394
aataataata ataataataa 20
<210> 395
<211> 20
<212> DNA
<213> Homo sapiens
<400> 395
ataataataa taataataat 20
<210> 396
<211> 20
<212> DNA
<213> Homo sapiens
<400> 396
taataataat aataataata 20
<210> 397
<211> 20
<212> DNA
<213> Homo sapiens
<400> 397
aataataata ataataataa 20
<210> 398
<211> 20
<212> DNA
<213> Homo sapiens
<400> 398
ataataataa taataataaa 20
<210> 399
<211> 20
<212> DNA
<213> Homo sapiens
<400> 399
taataataat aataataaaa 20
<210> 400
<211> 20
<212> DNA
<213> Homo sapiens
<400> 400
aataataata ataataaaag 20
<210> 401
<211> 20
<212> DNA
<213> Homo sapiens
<400> 401
ataataataa taataaaagc 20
<210> 402
<211> 20
<212> DNA
<213> Homo sapiens
<400> 402
taataataat aataaaagca 20
<210> 403
<211> 20
<212> DNA
<213> Homo sapiens
<400> 403
aataataata ataaaagcag 20
<210> 404
<211> 20
<212> DNA
<213> Homo sapiens
<400> 404
ataataataa taaaagcagt 20
<210> 405
<211> 20
<212> DNA
<213> Homo sapiens
<400> 405
taataataat aaaagcagtt 20
<210> 406
<211> 20
<212> DNA
<213> Homo sapiens
<400> 406
aaaattaaaa aaaatcatta 20
<210> 407
<211> 20
<212> DNA
<213> Homo sapiens
<400> 407
aaattaaaaa aaatcattaa 20
<210> 408
<211> 20
<212> DNA
<213> Homo sapiens
<400> 408
aattaaaaaa aatcattaaa 20
<210> 409
<211> 20
<212> DNA
<213> Homo sapiens
<400> 409
attaaaaaaa atcattaaaa 20
<210> 410
<211> 20
<212> DNA
<213> Homo sapiens
<400> 410
ttaaaaaaaa tcattaaaac 20
<210> 411
<211> 20
<212> DNA
<213> Homo sapiens
<400> 411
taaaaaaaat cattaaaact 20
<210> 412
<211> 20
<212> DNA
<213> Homo sapiens
<400> 412
aaaaaaaatc attaaaacta 20
<210> 413
<211> 20
<212> DNA
<213> Homo sapiens
<400> 413
aaaaaaatca ttaaaactat 20
<210> 414
<211> 20
<212> DNA
<213> Homo sapiens
<400> 414
aaaaaatcat taaaactatg 20
<210> 415
<211> 20
<212> DNA
<213> Homo sapiens
<400> 415
aaaaatcatt aaaactatgt 20
<210> 416
<211> 20
<212> DNA
<213> Homo sapiens
<400> 416
aaaatcatta aaactatgtt 20
<210> 417
<211> 20
<212> DNA
<213> Homo sapiens
<400> 417
aaatcattaa aactatgttc 20
<210> 418
<211> 20
<212> DNA
<213> Homo sapiens
<400> 418
aatcattaaa actatgttca 20
<210> 419
<211> 20
<212> DNA
<213> Homo sapiens
<400> 419
atcattaaaa ctatgttcat 20
<210> 420
<211> 20
<212> DNA
<213> Homo sapiens
<400> 420
tcattaaaac tatgttcatt 20
<210> 421
<211> 20
<212> DNA
<213> Homo sapiens
<400> 421
cattaaaact atgttcattt 20
<210> 422
<211> 20
<212> DNA
<213> Homo sapiens
<400> 422
attaaaacta tgttcatttt 20
<210> 423
<211> 20
<212> DNA
<213> Homo sapiens
<400> 423
ttaaaactat gttcatttta 20
<210> 424
<211> 20
<212> DNA
<213> Homo sapiens
<400> 424
taaaactatg ttcattttac 20
<210> 425
<211> 20
<212> DNA
<213> Homo sapiens
<400> 425
aaaactatgt tcattttact 20
<210> 426
<211> 20
<212> DNA
<213> Homo sapiens
<400> 426
aaactatgtt cattttactt 20
<210> 427
<211> 20
<212> DNA
<213> Homo sapiens
<400> 427
aactatgttc attttacttt 20
<210> 428
<211> 20
<212> DNA
<213> Homo sapiens
<400> 428
actatgttca ttttactttc 20
<210> 429
<211> 20
<212> DNA
<213> Homo sapiens
<400> 429
ctatgttcat tttactttca 20
<210> 430
<211> 20
<212> DNA
<213> Homo sapiens
<400> 430
caagttagat aatataaaag 20
<210> 431
<211> 20
<212> DNA
<213> Homo sapiens
<400> 431
aagttagata atataaaagc 20
<210> 432
<211> 20
<212> DNA
<213> Homo sapiens
<400> 432
agttagataa tataaaagct 20
<210> 433
<211> 20
<212> DNA
<213> Homo sapiens
<400> 433
gttagataat ataaaagcta 20
<210> 434
<211> 20
<212> DNA
<213> Homo sapiens
<400> 434
ttagataata taaaagctag 20
<210> 435
<211> 20
<212> DNA
<213> Homo sapiens
<400> 435
tttggtggtt tttcacactt 20
<210> 436
<211> 20
<212> DNA
<213> Homo sapiens
<400> 436
ttggtggttt ttcacactta 20
<210> 437
<211> 20
<212> DNA
<213> Homo sapiens
<400> 437
tggtggtttt tcacacttat 20
<210> 438
<211> 20
<212> DNA
<213> Homo sapiens
<400> 438
ggtggttttt cacacttatc 20
<210> 439
<211> 20
<212> DNA
<213> Homo sapiens
<400> 439
gtggtttttc acacttatca 20
<210> 440
<211> 20
<212> DNA
<213> Homo sapiens
<400> 440
tggtttttca cacttatcat 20
<210> 441
<211> 20
<212> DNA
<213> Homo sapiens
<400> 441
ggtttttcac acttatcatc 20
<210> 442
<211> 20
<212> DNA
<213> Homo sapiens
<400> 442
gtttttcaca cttatcatca 20
<210> 443
<211> 20
<212> DNA
<213> Homo sapiens
<400> 443
tttttcacac ttatcatcat 20
<210> 444
<211> 20
<212> DNA
<213> Homo sapiens
<400> 444
ttttcacact tatcatcatt 20
<210> 445
<211> 20
<212> DNA
<213> Homo sapiens
<400> 445
tttcacactt atcatcattt 20
<210> 446
<211> 20
<212> DNA
<213> Homo sapiens
<400> 446
ttcacactta tcatcatttc 20
<210> 447
<211> 20
<212> DNA
<213> Homo sapiens
<400> 447
tcacacttat catcatttct 20
<210> 448
<211> 20
<212> DNA
<213> Homo sapiens
<400> 448
cacacttatc atcatttctt 20
<210> 449
<211> 20
<212> DNA
<213> Homo sapiens
<400> 449
acacttatca tcatttcttc 20
<210> 450
<211> 20
<212> DNA
<213> Homo sapiens
<400> 450
cacttatcat catttcttcg 20
<210> 451
<211> 20
<212> DNA
<213> Homo sapiens
<400> 451
acttatcatc atttcttcgt 20
<210> 452
<211> 20
<212> DNA
<213> Homo sapiens
<400> 452
cttatcatca tttcttcgta 20
<210> 453
<211> 20
<212> DNA
<213> Homo sapiens
<400> 453
ttatcatcat ttcttcgtat 20
<210> 454
<211> 20
<212> DNA
<213> Homo sapiens
<400> 454
tatcatcatt tcttcgtata 20
<210> 455
<211> 20
<212> DNA
<213> Homo sapiens
<400> 455
atcatcattt cttcgtatac 20
<210> 456
<211> 20
<212> DNA
<213> Homo sapiens
<400> 456
tcatcatttc ttcgtatact 20
<210> 457
<211> 20
<212> DNA
<213> Homo sapiens
<400> 457
catcatttct tcgtatactg 20
<210> 458
<211> 20
<212> DNA
<213> Homo sapiens
<400> 458
atcatttctt cgtatactgc 20
<210> 459
<211> 20
<212> DNA
<213> Homo sapiens
<400> 459
tcatttcttc gtatactgct 20
<210> 460
<211> 20
<212> DNA
<213> Homo sapiens
<400> 460
catttcttcg tatactgcta 20
<210> 461
<211> 20
<212> DNA
<213> Homo sapiens
<400> 461
atttcttcgt atactgctaa 20
<210> 462
<211> 20
<212> DNA
<213> Homo sapiens
<400> 462
tttcttcgta tactgctaac 20
<210> 463
<211> 20
<212> DNA
<213> Homo sapiens
<400> 463
ttcttcgtat actgctaact 20
<210> 464
<211> 20
<212> DNA
<213> Homo sapiens
<400> 464
tcttcgtata ctgctaactt 20
<210> 465
<211> 20
<212> DNA
<213> Homo sapiens
<400> 465
cttcgtatac tgctaactta 20
<210> 466
<211> 20
<212> DNA
<213> Homo sapiens
<400> 466
ttcgtatact gctaacttag 20
<210> 467
<211> 20
<212> DNA
<213> Homo sapiens
<400> 467
tcgtatactg ctaacttagc 20
<210> 468
<211> 20
<212> DNA
<213> Homo sapiens
<400> 468
cgtatactgc taacttagcc 20
<210> 469
<211> 20
<212> DNA
<213> Homo sapiens
<400> 469
gtatactgct aacttagccg 20
<210> 470
<211> 20
<212> DNA
<213> Homo sapiens
<400> 470
tatactgcta acttagccgc 20
<210> 471
<211> 20
<212> DNA
<213> Homo sapiens
<400> 471
atactgctaa cttagccgcc 20
<210> 472
<211> 20
<212> DNA
<213> Homo sapiens
<400> 472
tactgctaac ttagccgcct 20
<210> 473
<211> 20
<212> DNA
<213> Homo sapiens
<400> 473
actgctaact tagccgcctt 20
<210> 474
<211> 20
<212> DNA
<213> Homo sapiens
<400> 474
ctgctaactt agccgccttt 20
<210> 475
<211> 20
<212> DNA
<213> Homo sapiens
<400> 475
tgctaactta gccgcctttc 20
<210> 476
<211> 20
<212> DNA
<213> Homo sapiens
<400> 476
gctaacttag ccgcctttct 20
<210> 477
<211> 20
<212> DNA
<213> Homo sapiens
<400> 477
ctaacttagc cgcctttctg 20
<210> 478
<211> 20
<212> DNA
<213> Homo sapiens
<400> 478
taacttagcc gcctttctga 20
<210> 479
<211> 20
<212> DNA
<213> Homo sapiens
<400> 479
aacttagccg cctttctgac 20
<210> 480
<211> 20
<212> DNA
<213> Homo sapiens
<400> 480
acttagccgc ctttctgaca 20
<210> 481
<211> 20
<212> DNA
<213> Homo sapiens
<400> 481
cttagccgcc tttctgacag 20
<210> 482
<211> 20
<212> DNA
<213> Homo sapiens
<400> 482
ttagccgcct ttctgacagt 20
<210> 483
<211> 20
<212> DNA
<213> Homo sapiens
<400> 483
tagccgcctt tctgacagtg 20
<210> 484
<211> 20
<212> DNA
<213> Homo sapiens
<400> 484
agccgccttt ctgacagtgg 20
<210> 485
<211> 20
<212> DNA
<213> Homo sapiens
<400> 485
gccgcctttc tgacagtgga 20
<210> 486
<211> 20
<212> DNA
<213> Homo sapiens
<400> 486
ccgcctttct gacagtggaa 20
<210> 487
<211> 20
<212> DNA
<213> Homo sapiens
<400> 487
cgcctttctg acagtggaac 20
<210> 488
<211> 20
<212> DNA
<213> Homo sapiens
<400> 488
gcctttctga cagtggaacg 20
<210> 489
<211> 20
<212> DNA
<213> Homo sapiens
<400> 489
cctttctgac agtggaacgc 20
<210> 490
<211> 20
<212> DNA
<213> Homo sapiens
<400> 490
ctttctgaca gtggaacgca 20
<210> 491
<211> 20
<212> DNA
<213> Homo sapiens
<400> 491
tttctgacag tggaacgcat 20
<210> 492
<211> 20
<212> DNA
<213> Homo sapiens
<400> 492
ttctgacagt ggaacgcatg 20
<210> 493
<211> 20
<212> DNA
<213> Homo sapiens
<400> 493
tctgacagtg gaacgcatgg 20
<210> 494
<211> 20
<212> DNA
<213> Homo sapiens
<400> 494
ctgacagtgg aacgcatgga 20
<210> 495
<211> 20
<212> DNA
<213> Homo sapiens
<400> 495
ggtgcaacca tgactttttt 20
<210> 496
<211> 20
<212> DNA
<213> Homo sapiens
<400> 496
gtgcaaccat gacttttttc 20
<210> 497
<211> 20
<212> DNA
<213> Homo sapiens
<400> 497
tgcaaccatg acttttttca 20
<210> 498
<211> 20
<212> DNA
<213> Homo sapiens
<400> 498
gcaaccatga cttttttcaa 20
<210> 499
<211> 20
<212> DNA
<213> Homo sapiens
<400> 499
caaccatgac ttttttcaag 20
<210> 500
<211> 20
<212> DNA
<213> Homo sapiens
<400> 500
aaccatgact tttttcaaga 20
<210> 501
<211> 20
<212> DNA
<213> Homo sapiens
<400> 501
accatgactt ttttcaagaa 20
<210> 502
<211> 20
<212> DNA
<213> Homo sapiens
<400> 502
ccatgacttt tttcaagaaa 20
<210> 503
<211> 20
<212> DNA
<213> Homo sapiens
<400> 503
catgactttt ttcaagaaat 20
<210> 504
<211> 20
<212> DNA
<213> Homo sapiens
<400> 504
atgacttttt tcaagaaatc 20
<210> 505
<211> 20
<212> DNA
<213> Homo sapiens
<400> 505
tgactttttt caagaaatca 20
<210> 506
<211> 20
<212> DNA
<213> Homo sapiens
<400> 506
gacttttttc aagaaatcaa 20
<210> 507
<211> 20
<212> DNA
<213> Homo sapiens
<400> 507
acttttttca agaaatcaaa 20
<210> 508
<211> 20
<212> DNA
<213> Homo sapiens
<400> 508
cttttttcaa gaaatcaaaa 20
<210> 509
<211> 20
<212> DNA
<213> Homo sapiens
<400> 509
ttttttcaag aaatcaaaaa 20
<210> 510
<211> 20
<212> DNA
<213> Homo sapiens
<400> 510
tttttcaaga aatcaaaaat 20
<210> 511
<211> 20
<212> DNA
<213> Homo sapiens
<400> 511
ttttcaagaa atcaaaaatc 20
<210> 512
<211> 20
<212> DNA
<213> Homo sapiens
<400> 512
tttcaagaaa tcaaaaatct 20
<210> 513
<211> 20
<212> DNA
<213> Homo sapiens
<400> 513
aggttccttt tgaagctcaa 20
<210> 514
<211> 20
<212> DNA
<213> Homo sapiens
<400> 514
ggttcctttt gaagctcaac 20
<210> 515
<211> 20
<212> DNA
<213> Homo sapiens
<400> 515
gttccttttg aagctcaact 20
<210> 516
<211> 20
<212> DNA
<213> Homo sapiens
<400> 516
ttccttttga agctcaactg 20
<210> 517
<211> 20
<212> DNA
<213> Homo sapiens
<400> 517
tccttttgaa gctcaactgt 20
<210> 518
<211> 20
<212> DNA
<213> Homo sapiens
<400> 518
ccttttgaag ctcaactgtt 20
<210> 519
<211> 20
<212> DNA
<213> Homo sapiens
<400> 519
cttttgaagc tcaactgttg 20
<210> 520
<211> 20
<212> DNA
<213> Homo sapiens
<400> 520
ttttgaagct caactgttgc 20
<210> 521
<211> 20
<212> DNA
<213> Homo sapiens
<400> 521
tttgaagctc aactgttgcc 20
<210> 522
<211> 20
<212> DNA
<213> Homo sapiens
<400> 522
ttgaagctca actgttgcca 20
<210> 523
<211> 20
<212> DNA
<213> Homo sapiens
<400> 523
tgaagctcaa ctgttgccag 20
<210> 524
<211> 20
<212> DNA
<213> Homo sapiens
<400> 524
gaagctcaac tgttgccagg 20
<210> 525
<211> 20
<212> DNA
<213> Homo sapiens
<400> 525
aagctcaact gttgccagga 20
<210> 526
<211> 20
<212> DNA
<213> Homo sapiens
<400> 526
agctcaactg ttgccaggag 20
<210> 527
<211> 20
<212> DNA
<213> Homo sapiens
<400> 527
gctcaactgt tgccaggaga 20
<210> 528
<211> 20
<212> DNA
<213> Homo sapiens
<400> 528
ctcaactgtt gccaggagat 20
<210> 529
<211> 20
<212> DNA
<213> Homo sapiens
<400> 529
tcaactgttg ccaggagatg 20
<210> 530
<211> 20
<212> DNA
<213> Homo sapiens
<400> 530
caactgttgc caggagatgg 20
<210> 531
<211> 20
<212> DNA
<213> Homo sapiens
<400> 531
aactgttgcc aggagatgga 20
<210> 532
<211> 20
<212> DNA
<213> Homo sapiens
<400> 532
actgttgcca ggagatggaa 20
<210> 533
<211> 20
<212> DNA
<213> Homo sapiens
<400> 533
ctgttgccag gagatggaat 20
<210> 534
<211> 20
<212> DNA
<213> Homo sapiens
<400> 534
tgttgccagg agatggaata 20
<210> 535
<211> 20
<212> DNA
<213> Homo sapiens
<400> 535
gttgccagga gatggaatat 20
<210> 536
<211> 20
<212> DNA
<213> Homo sapiens
<400> 536
ttgccaggag atggaatatc 20
<210> 537
<211> 20
<212> DNA
<213> Homo sapiens
<400> 537
tgccaggaga tggaatatca 20
<210> 538
<211> 20
<212> DNA
<213> Homo sapiens
<400> 538
gccaggagat ggaatatcaa 20
<210> 539
<211> 20
<212> DNA
<213> Homo sapiens
<400> 539
catggcagtt ggaatataaa 20
<210> 540
<211> 20
<212> DNA
<213> Homo sapiens
<400> 540
atggcagttg gaatataaag 20
<210> 541
<211> 20
<212> DNA
<213> Homo sapiens
<400> 541
tggcagttgg aatataaagc 20
<210> 542
<211> 20
<212> DNA
<213> Homo sapiens
<400> 542
ggcagttgga atataaagca 20
<210> 543
<211> 20
<212> DNA
<213> Homo sapiens
<400> 543
gcagttggaa tataaagcag 20
<210> 544
<211> 20
<212> DNA
<213> Homo sapiens
<400> 544
cagttggaat ataaagcaga 20
<210> 545
<211> 20
<212> DNA
<213> Homo sapiens
<400> 545
agttggaata taaagcagat 20
<210> 546
<211> 20
<212> DNA
<213> Homo sapiens
<400> 546
gttggaatat aaagcagatg 20
<210> 547
<211> 20
<212> DNA
<213> Homo sapiens
<400> 547
ttggaatata aagcagatgt 20
<210> 548
<211> 20
<212> DNA
<213> Homo sapiens
<400> 548
tggaatataa agcagatgtt 20
<210> 549
<211> 20
<212> DNA
<213> Homo sapiens
<400> 549
ggaatataaa gcagatgttc 20
<210> 550
<211> 20
<212> DNA
<213> Homo sapiens
<400> 550
gaatataaag cagatgttca 20
<210> 551
<211> 20
<212> DNA
<213> Homo sapiens
<400> 551
aatataaagc agatgttcat 20
<210> 552
<211> 20
<212> DNA
<213> Homo sapiens
<400> 552
atataaagca gatgttcatc 20
<210> 553
<211> 20
<212> DNA
<213> Homo sapiens
<400> 553
tataaagcag atgttcatca 20
<210> 554
<211> 20
<212> DNA
<213> Homo sapiens
<400> 554
ataaagcaga tgttcatcac 20
<210> 555
<211> 20
<212> DNA
<213> Homo sapiens
<400> 555
taaagcagat gttcatcact 20
<210> 556
<211> 20
<212> DNA
<213> Homo sapiens
<400> 556
aaagcagatg ttcatcactt 20
<210> 557
<211> 20
<212> DNA
<213> Homo sapiens
<400> 557
aagcagatgt tcatcactta 20
<210> 558
<211> 20
<212> DNA
<213> Homo sapiens
<400> 558
agcagatgtt catcacttat 20
<210> 559
<211> 20
<212> DNA
<213> Homo sapiens
<400> 559
gcagatgttc atcacttatt 20
<210> 560
<211> 20
<212> DNA
<213> Homo sapiens
<400> 560
cagatgttca tcacttattt 20
<210> 561
<211> 20
<212> DNA
<213> Homo sapiens
<400> 561
agatgttcat cacttatttt 20
<210> 562
<211> 20
<212> DNA
<213> Homo sapiens
<400> 562
gatgttcatc acttattttc 20
<210> 563
<211> 20
<212> DNA
<213> Homo sapiens
<400> 563
atgttcatca cttattttcc 20
<210> 564
<211> 20
<212> DNA
<213> Homo sapiens
<400> 564
tgttcatcac ttattttcct 20
<210> 565
<211> 20
<212> DNA
<213> Homo sapiens
<400> 565
gttcatcact tattttcctt 20
<210> 566
<211> 20
<212> DNA
<213> Homo sapiens
<400> 566
ttcatcactt attttccttt 20
<210> 567
<211> 20
<212> DNA
<213> Homo sapiens
<400> 567
tcatcactta ttttcctttt 20
<210> 568
<211> 20
<212> DNA
<213> Homo sapiens
<400> 568
catcacttat tttccttttt 20
<210> 569
<211> 20
<212> DNA
<213> Homo sapiens
<400> 569
atcacttatt ttcctttttt 20
<210> 570
<211> 20
<212> DNA
<213> Homo sapiens
<400> 570
tcacttattt tccttttttc 20
<210> 571
<211> 20
<212> DNA
<213> Homo sapiens
<400> 571
cacttatttt ccttttttct 20
<210> 572
<211> 20
<212> DNA
<213> Homo sapiens
<400> 572
acttattttc cttttttctt 20
<210> 573
<211> 20
<212> DNA
<213> Homo sapiens
<400> 573
atgccaatta tcaaggaaat 20
<210> 574
<211> 20
<212> DNA
<213> Homo sapiens
<400> 574
tgccaattat caaggaaata 20
<210> 575
<211> 20
<212> DNA
<213> Homo sapiens
<400> 575
gccaattatc aaggaaataa 20
<210> 576
<211> 20
<212> DNA
<213> Homo sapiens
<400> 576
ccaattatca aggaaataat 20
<210> 577
<211> 20
<212> DNA
<213> Homo sapiens
<400> 577
caattatcaa ggaaataatg 20
<210> 578
<211> 20
<212> DNA
<213> Homo sapiens
<400> 578
aattatcaag gaaataatgt 20
<210> 579
<211> 20
<212> DNA
<213> Homo sapiens
<400> 579
attatcaagg aaataatgtt 20
<210> 580
<211> 20
<212> DNA
<213> Homo sapiens
<400> 580
ttatcaagga aataatgttt 20
<210> 581
<211> 20
<212> DNA
<213> Homo sapiens
<400> 581
tatcaaggaa ataatgtttt 20
<210> 582
<211> 21
<212> DNA
<213> Homo sapiens
<400> 582
ttgctaccca atactaccct t 21
<210> 583
<211> 21
<212> DNA
<213> Homo sapiens
<400> 583
gtggctgcca tcttcgggcc t 21
<210> 584
<211> 21
<212> DNA
<213> Homo sapiens
<400> 584
gtgcagtcca tctgcaatgc t 21
<210> 585
<211> 21
<212> DNA
<213> Homo sapiens
<400> 585
accgatacag tggtgttaac a 21
<210> 586
<211> 21
<212> DNA
<213> Homo sapiens
<400> 586
aggcacctcc gaaacccgat t 21
<210> 587
<211> 21
<212> DNA
<213> Homo sapiens
<400> 587
caggcacctc cgaaacccga t 21
<210> 588
<211> 21
<212> DNA
<213> Homo sapiens
<400> 588
ctcctcagag agttatctac a 21
<210> 589
<211> 21
<212> DNA
<213> Homo sapiens
<400> 589
atggaatggt tcgtgaacta a 21
<210> 590
<211> 21
<212> DNA
<213> Homo sapiens
<400> 590
aagccacagg ccccagttat t 21
<210> 591
<211> 21
<212> DNA
<213> Homo sapiens
<400> 591
aaccaataaa agtgtcacta a 21
<210> 592
<211> 21
<212> DNA
<213> Homo sapiens
<400> 592
cccccatatc ccaaataata a 21
<210> 593
<211> 21
<212> DNA
<213> Homo sapiens
<400> 593
aagggaaagc cagcgaacat c 21
<210> 594
<211> 20
<212> DNA
<213> Homo sapiens
<400> 594
cgcctttctg acagtggaac 20
<210> 595
<211> 21
<212> DNA
<213> Homo sapiens
<400> 595
cgcctttctg acagtggaac g 21
<210> 596
<211> 21
<212> DNA
<213> Homo sapiens
<400> 596
ttgccaggag atggaatatc a 21
<210> 597
<211> 21
<212> DNA
<213> Homo sapiens
<400> 597
aagcagatgt tcatcactta t 21
<210> 598
<211> 21
<212> DNA
<213> Homo sapiens
<400> 598
atgccaatta tcaaggaaat a 21
<210> 599
<211> 20
<212> DNA
<213> Homo sapiens
<400> 599
atggtagaag aattgaggat 20
<210> 600
<211> 21
<212> DNA
<213> Homo sapiens
<400> 600
atggtagaag aattgaggat g 21
<210> 601
<211> 21
<212> DNA
<213> Homo sapiens
<400> 601
ttccttgcag tgtaatcgac a 21
<210> 602
<211> 21
<212> DNA
<213> Homo sapiens
<400> 602
gcggtcacca ctcgacgcat c 21
<210> 603
<211> 21
<212> DNA
<213> Homo sapiens
<400> 603
tggctattac ctatgttcga g 21
<210> 604
<211> 21
<212> DNA
<213> Homo sapiens
<400> 604
atcctcattt ctacccgaac c 21
<210> 605
<211> 20
<212> DNA
<213> Homo sapiens
<400> 605
aacccaggag ccgaacgcta 20
<210> 606
<211> 21
<212> DNA
<213> Homo sapiens
<400> 606
aacccaggag ccgaacgcta g 21
<210> 607
<211> 21
<212> DNA
<213> Homo sapiens
<400> 607
acccgaaccc aggagccgaa c 21
<210> 608
<211> 21
<212> DNA
<213> Homo sapiens
<400> 608
tgcgtgtgca ccaccaacgt t 21
<210> 609
<211> 21
<212> DNA
<213> Homo sapiens
<400> 609
atccagtctt caggcgcacc a 21
<210> 610
<211> 21
<212> DNA
<213> Homo sapiens
<400> 610
atccagtctt caggcgcacc g 21
<210> 611
<211> 20
<212> DNA
<213> Homo sapiens
<400> 611
tccccacata cagacccgct 20
<210> 612
<211> 21
<212> DNA
<213> Homo sapiens
<400> 612
tccccacata cagacccgct g 21
<210> 613
<211> 20
<212> DNA
<213> Homo sapiens
<400> 613
aagcatgaat ggtagtgcgt 20
<210> 614
<211> 21
<212> DNA
<213> Homo sapiens
<400> 614
aagcatgaat ggtagtgcgt g 21
<210> 615
<211> 20
<212> DNA
<213> Homo sapiens
<400> 615
atggagtcaa caaccatcga 20
<210> 616
<211> 21
<212> DNA
<213> Homo sapiens
<400> 616
atggagtcaa caaccatcga g 21
<210> 617
<211> 21
<212> DNA
<213> Homo sapiens
<400> 617
ttccccacat acagacccgc t 21
<210> 618
<211> 21
<212> DNA
<213> Homo sapiens
<400> 618
gtggtgtctg tggccgttca a 21
<210> 619
<211> 21
<212> DNA
<213> Homo sapiens
<400> 619
tggcgcttcg ggacccgctt t 21
<210> 620
<211> 21
<212> DNA
<213> Homo sapiens
<400> 620
atggtacaaa cccaggcgtc t 21
<210> 621
<211> 21
<212> DNA
<213> Homo sapiens
<400> 621
cagcactggt ctcattcgtt t 21
<210> 622
<211> 21
<212> DNA
<213> Homo sapiens
<400> 622
ttggaagtgg actgcgttta t 21
<210> 623
<211> 21
<212> DNA
<213> Homo sapiens
<400> 623
gccgtgatcg tgtctgcggt c 21
<210> 624
<211> 21
<212> DNA
<213> Homo sapiens
<400> 624
tacccgaacc caggagccga a 21
<210> 625
<211> 21
<212> DNA
<213> Homo sapiens
<400> 625
ggcgcttcgg gacccgcttt a 21
<210> 626
<211> 20
<212> DNA
<213> Homo sapiens
<400> 626
atcggggaag tgggtgccgt 20
<210> 627
<211> 21
<212> DNA
<213> Homo sapiens
<400> 627
atcggggaag tgggtgccgt g 21
<210> 628
<211> 21
<212> DNA
<213> Homo sapiens
<400> 628
atcgacataa accctggcgc t 21
<210> 629
<211> 21
<212> DNA
<213> Homo sapiens
<400> 629
atgggtttgg gaagcggaga c 21
<210> 630
<211> 21
<212> DNA
<213> Homo sapiens
<400> 630
acccaagcac aaactgtcgt c 21
<210> 631
<211> 21
<212> DNA
<213> Homo sapiens
<400> 631
acccctggtt tccacgaggc t 21
<210> 632
<211> 21
<212> DNA
<213> Homo sapiens
<400> 632
tgggttctcc atatcgagac a 21
<210> 633
<211> 21
<212> DNA
<213> Homo sapiens
<400> 633
gtccctgaag tgccagcgtc a 21
<210> 634
<211> 21
<212> DNA
<213> Homo sapiens
<400> 634
gtccctgaag tgccagcgtc g 21
<210> 635
<211> 21
<212> DNA
<213> Homo sapiens
<400> 635
gaccatgccg tgatcgtgtc t 21
<210> 636
<211> 21
<212> DNA
<213> Homo sapiens
<400> 636
gccgaacgct agatcgggga a 21
<210> 637
<211> 21
<212> DNA
<213> Homo sapiens
<400> 637
gtggtcgatg gaacgattgc a 21
<210> 638
<211> 20
<212> DNA
<213> Homo sapiens
<400> 638
cacctccgaa acccgattca 20
<210> 639
<211> 21
<212> DNA
<213> Homo sapiens
<400> 639
cacctccgaa acccgattca g 21
<210> 640
<211> 21
<212> DNA
<213> Homo sapiens
<400> 640
tggctgccat cttcgggcct t 21
<210> 641
<211> 21
<212> DNA
<213> Homo sapiens
<400> 641
aaggtataat cttcgactca a 21
<210> 642
<211> 21
<212> DNA
<213> Homo sapiens
<400> 642
gaggtgatga tgttactagc c 21
<210> 643
<211> 21
<212> DNA
<213> Homo sapiens
<400> 643
atcctctctc ccctgatatc t 21
<210> 644
<211> 21
<212> DNA
<213> Homo sapiens
<400> 644
ttcctgtgga aatatgcaac c 21
<210> 645
<211> 21
<212> DNA
<213> Homo sapiens
<400> 645
ctgcggtcac cactcgacgc a 21
<210> 646
<211> 21
<212> DNA
<213> Homo sapiens
<400> 646
tcccggatgc tctccgacta a 21
<210> 647
<211> 21
<212> DNA
<213> Homo sapiens
<400> 647
tgcggtcacc actcgacgca t 21
<210> 648
<211> 20
<212> DNA
<213> Homo sapiens
<400> 648
gtgctggatt tctcccggat 20
<210> 649
<211> 21
<212> DNA
<213> Homo sapiens
<400> 649
aaaacaggca ttagctatgg g 21
<210> 650
<211> 21
<212> DNA
<213> Homo sapiens
<400> 650
tagctatggg aatgatgaca a 21
<210> 651
<211> 21
<212> DNA
<213> Homo sapiens
<400> 651
caggcattag ctatgggaat g 21
<210> 652
<211> 20
<212> DNA
<213> Homo sapiens
<400> 652
aaagcaggca ttagctatgg 20
<210> 653
<211> 21
<212> DNA
<213> Homo sapiens
<400> 653
gtgggaggca tttggtggtt t 21
<210> 654
<211> 21
<212> DNA
<213> Homo sapiens
<400> 654
gtggtttttc acacttatca t 21
<210> 655
<211> 21
<212> DNA
<213> Homo sapiens
<400> 655
ttcgagagaa ggtcatcgac t 21
<210> 656
<211> 20
<212> DNA
<213> Homo sapiens
<400> 656
tggctattac ctatgttcga 20
<210> 657
<211> 21
<212> DNA
<213> Homo sapiens
<400> 657
ccgctggaag caccaggtgt c 21
<210> 658
<211> 21
<212> DNA
<213> Homo sapiens
<400> 658
atgggttctc catatcgaga c 21
<210> 659
<211> 21
<212> DNA
<213> Homo sapiens
<400> 659
tcccatgggt tctccatatc a 21
<210> 660
<211> 21
<212> DNA
<213> Homo sapiens
<400> 660
ctcccatggg ttctccatat c 21
<210> 661
<211> 21
<212> DNA
<213> Homo sapiens
<400> 661
cagtccatct gcaatgctct g 21
<210> 662
<211> 21
<212> DNA
<213> Homo sapiens
<400> 662
gtccatctgc aatgctctgg g 21
<210> 663
<211> 20
<212> DNA
<213> Homo sapiens
<400> 663
aaccctgact cagacgtggt 20
<210> 664
<211> 21
<212> DNA
<213> Homo sapiens
<400> 664
aaccctgact cagacgtggt g 21
<210> 665
<211> 21
<212> DNA
<213> Homo sapiens
<400> 665
acccttgcaa ccctgactca g 21
<210> 666
<211> 21
<212> DNA
<213> Homo sapiens
<400> 666
ctggtttgga gttggagctc t 21
<210> 667
<211> 21
<212> DNA
<213> Homo sapiens
<400> 667
gagctcatca aagctccatc a 21
<210> 668
<211> 20
<212> DNA
<213> Homo sapiens
<400> 668
ctccactggc tattacctat 20
<210> 669
<211> 21
<212> DNA
<213> Homo sapiens
<400> 669
ctccactggc tattacctat g 21
<210> 670
<211> 21
<212> DNA
<213> Homo sapiens
<400> 670
aagctgacct tgcagttgct c 21
<210> 671
<211> 21
<212> DNA
<213> Homo sapiens
<400> 671
ttgctccact ggctattacc t 21
<210> 672
<211> 21
<212> DNA
<213> Homo sapiens
<400> 672
ctggatggat ttatgacgac t 21
<210> 673
<211> 21
<212> DNA
<213> Homo sapiens
<400> 673
aaagattcct tctatgtcag t 21
<210> 674
<211> 21
<212> DNA
<213> Homo sapiens
<400> 674
ttccttctat gtcagtctct a 21
<210> 675
<211> 21
<212> DNA
<213> Homo sapiens
<400> 675
ttgctggatg gatttatgac a 21
<210> 676
<211> 21
<212> DNA
<213> Homo sapiens
<400> 676
atggatttat gacgactgat a 21
<210> 677
<211> 21
<212> DNA
<213> Homo sapiens
<400> 677
atccttggct ttacatatga a 21
<210> 678
<211> 21
<212> DNA
<213> Homo sapiens
<400> 678
aaggattcct tctatgtcag t 21
<210> 679
<211> 21
<212> DNA
<213> Homo sapiens
<400> 679
cattccattc cattgtccat t 21
<210> 680
<211> 21
<212> DNA
<213> Homo sapiens
<400> 680
tcacgaacca ttccattcca t 21
<210> 681
<211> 21
<212> DNA
<213> Homo sapiens
<400> 681
ctcccggatg ctctccgact a 21
<210> 682
<211> 429
<212> DNA
<213> Homo sapiens
<400> 682
ctgcagaggg ccctgcgtat gagtgcaagt gggttttagg accaggatga ggcggggtgg 60
gggtgcctac ctgacgaccg accccgaccc actggacaag cacccaaccc ccattcccca 120
aattgcgcat cccctatcag agagggggag gggaaacagg atgcggcgag gcgcgtgcgc 180
actgccagct tcagcaccgc ggacagtgcc ttcgcccccg cctggcggcg cgcgccaccg 240
ccgcctcagc actgaaggcg cgctgacgtc actcgccggt cccccgcaaa ctccccttcc 300
cggccacctt ggtcgcgtcc gcgccgccgc cggcccagcc ggaccgcacc acgcgaggcg 360
cgagataggg gggcacgggc gcgaccatct gcgctgcggc gccggcgact cagcgctgcc 420
tcagtctgc 429
<210> 683
<211> 469
<212> DNA
<213> Homo sapiens
<400> 683
ctgcagaggg ccctgcgtat gagtgcaagt gggttttagg accaggatga ggcggggtgg 60
gggtgcctac ctgacgaccg accccgaccc actggacaag cacccaaccc ccattcccca 120
aattgcgcat cccctatcag agagggggag gggaaacagg atgcggcgag gcgcgtgcgc 180
actgccagct tcagcaccgc ggacagtgcc ttcgcccccg cctggcggcg cgcgccaccg 240
ccgcctcagc actgaaggcg cgctgacgtc actcgccggt cccccgcaaa ctccccttcc 300
cggccacctt ggtcgcgtcc gcgccgccgc cggcccagcc ggaccgcacc acgcgaggcg 360
cgagataggg gggcacgggc gcgaccatct gcgctgcggc gccggcgact cagcgctgcc 420
tcagtctgcc aattgcagcg gaggagtcgt gtcgtgcctg agagcgcag 469
<210> 684
<211> 401
<212> DNA
<213> Homo sapiens
<400> 684
ctgcagaggg ccctgcgtat gagtgcaagt gggttttagg accaggatga ggcggggtgg 60
gggtgcctac ctgacgaccg accccgaccc actggacaag cacccaaccc ccattcccca 120
aattgcgcat cccctatcag agagggggag gggaaacagg atgcggcgag gcgcgtgcgc 180
actgccagct tcagcaccgc ggacagtgcc ttcgcccccg cctggcggcg cgcgccaccg 240
ccgcctcagc actgaaggcg cgctgacgtc actcgccggt cccccgcaaa ctccccttcc 300
cggccacctt ggtcgcgtcc gcgccgccgc cggcccagcc ggaccgcacc acgcgaggcg 360
cgagataggg gggcacgggc gcgaccatct gcgctgcggc g 401
<210> 685
<211> 477
<212> DNA
<213> Homo sapiens
<400> 685
ctgcagaggg ccctgcgtat gagtgcaagt gggttttagg accaggatga ggcggggtgg 60
gggtgcctac ctgacgaccg accccgaccc actggacaag cacccaaccc ccattcccca 120
aattgcgcat cccctatcag agagggggag gggaaacagg atgcggcgag gcgcgtgcgc 180
actgccagct tcagcaccgc ggacagtgcc ttcgcccccg cctggcggcg cgcgccaccg 240
ccgcctcagc actgaaggcg cgctgacgtc actcgccggt cccccgcaaa ctccccttcc 300
cggccacctt ggtcgcgtcc gcgccgccgc cggcccagcc ggaccgcacc acgcgaggcg 360
cgagataggg gggcacgggc gcgaccatct gcgctgcggc gccggcgact cagcgctgcc 420
tcagtctgcg gtgggcagcg gaggagtcgt gtcgtgcctg agagcgcagg gcgcgcc 477
<210> 686
<211> 60
<212> DNA
<213> Homo sapiens
<400> 686
gaggaggagg agagagaccg ggagggcgcc cgggaggcag ggcgcgcgca cactccgagg 60
<210> 687
<211> 60
<212> DNA
<213> Homo sapiens
<400> 687
gatgctgacg aaggctcgcg aggctgtgag cagccacagt gccctgctca gaagccccgg 60
<210> 688
<211> 60
<212> DNA
<213> Homo sapiens
<400> 688
gtctcccgcg cccgcgcccg tgtcgccgcc gtgcccgcga gcgggagccg gagtcgccgc 60
<210> 689
<211> 60
<212> DNA
<213> Homo sapiens
<400> 689
cgtgtgcaga tgcagggcgc cggtgccctg cgggtgcggg tgcaggagca gcgtgtgcag 60
<210> 690
<211> 60
<212> DNA
<213> Homo sapiens
<400> 690
ccccacgcca ccctttctgg tcatctcccc tcccgccccg cccctgcgca cactccctcg 60
<210> 691
<211> 60
<212> DNA
<213> Homo sapiens
<400> 691
tctccccggt aaagtctcgc ggtgctgccg ggctcagccc cgtctcctcc tcttgctccc 60
<210> 692
<211> 60
<212> DNA
<213> Homo sapiens
<400> 692
cgcctcctcc gcccgccgcc cgggagccgc agccgccgcc gccactgcca ctcccgctct 60
<210> 693
<211> 60
<212> DNA
<213> Homo sapiens
<400> 693
tgggtgcccc cacccttccc ccatcctcct cccttcccca ctccaccctc gtcggtcccc 60
<210> 694
<211> 60
<212> DNA
<213> Homo sapiens
<400> 694
aaaaaaaaaa aaaaagccca ccctccagcc tcgctgcaaa gagaaaaccg gagcagccgc 60
<210> 695
<211> 60
<212> DNA
<213> Homo sapiens
<400> 695
tctcgcactc tcccttctcc tttataaagg ccggaacagc tgaaagggtg gcaacttctc 60
<210> 696
<211> 60
<212> DNA
<213> Homo sapiens
<400> 696
gccgcgtcca cctgtcggcc gggcccagcc gagcgcgcag cgggcacgcc gcgcgcgcgg 60
<210> 697
<211> 60
<212> DNA
<213> Homo sapiens
<400> 697
gcgccccgcc cccggcgctg agtcctgtga cagcccccgg gccgcctgca cttgcagcct 60
<210> 698
<211> 60
<212> DNA
<213> Homo sapiens
<400> 698
aaagaagagt cccctattcc tgaaacttac tctgtccgtg gtgctgaaac attgtaccga 60
<210> 699
<211> 60
<212> DNA
<213> Homo sapiens
<400> 699
cgtcctcaaa gagcagcaag ccttctccat cttaatttga ctctaccgca gagcagactt 60
<210> 700
<211> 60
<212> DNA
<213> Homo sapiens
<400> 700
atgcagctat tctgttgtat tctcattctc actctccctc ccttctctca ctctcactct 60
<210> 701
<211> 60
<212> DNA
<213> Homo sapiens
<400> 701
catgttagcg tccccagctg cagcccaggg agggagagag gctgcgctca gtctgagagt 60
<210> 702
<211> 60
<212> DNA
<213> Homo sapiens
<400> 702
tgacgtcaga gagagagttt aaaacagagg gagacggttg agagcacaca agccgcttta 60
<210> 703
<211> 60
<212> DNA
<213> Homo sapiens
<400> 703
gagtgaaaat aaaagattgt ataaatcgtg gggcatgtgg aattgtgtgt gcctgtgcgt 60
<210> 704
<211> 60
<212> DNA
<213> Homo sapiens
<400> 704
gccgcggcga ggaagctcca taaaagccct gtcgcgaccc gctctctgca ccccatccgc 60
<210> 705
<211> 60
<212> DNA
<213> Homo sapiens
<400> 705
cagtcctaag tataagccct ataaaatgat gggctttgaa atgctggtca gggtagagtg 60
<210> 706
<211> 60
<212> DNA
<213> Homo sapiens
<400> 706
ttttccatta atgttttcag actgctgttg accacaggta actgaaatca tggaaagaga 60
<210> 707
<211> 60
<212> DNA
<213> Homo sapiens
<400> 707
tggtcatatg agcagaaatg atgagaaaag cactttttaa tcttttcgca cttgctctgc 60
<210> 708
<211> 60
<212> DNA
<213> Homo sapiens
<400> 708
aatagccaga gcagaagcct atataggtgg ccatcccacc tccaggctca cttcccgaca 60
<210> 709
<211> 60
<212> DNA
<213> Homo sapiens
<400> 709
cagcgctcag attttgcagc ataaatttgc atccaggaca gaccagagca gaggctgagg 60
<210> 710
<211> 60
<212> DNA
<213> Homo sapiens
<400> 710
gcacgcacgc gcgcgcaggg ccaagcccga ggcagctcgc ccgcagctcg cactcgcagg 60
<210> 711
<211> 60
<212> DNA
<213> Homo sapiens
<400> 711
cccgcctctg gctcgcccga ggacgcgctg gcacgcctcc caccccctca ctctgactcc 60
<210> 712
<211> 60
<212> DNA
<213> Homo sapiens
<400> 712
cactgggctc cctttccctc aaatgctctg gggctctccg cgctttcctg agtccgggct 60
<210> 713
<211> 60
<212> DNA
<213> Homo sapiens
<400> 713
cacagaaaac tcctctgggc cacgcttccc gcctcgccga ggtctcccca gtctgcccct 60
<210> 714
<211> 60
<212> DNA
<213> Homo sapiens
<400> 714
ctctgccccc gcctaccccg gagccgtgca gccgcctctc cgaatctctc tcttctcctg 60
<210> 715
<211> 60
<212> DNA
<213> Homo sapiens
<400> 715
ctggatttat aatcgcccta taaagctcca gaggcggtca ggcacctgca gaggagcccc 60
<210> 716
<211> 61
<212> DNA
<213> Homo sapiens
<400> 716
gggacgcgcg ggcggggtgg gctgtgcccc gcgggaaccc cgccggcctg tgcgcttgct 60
g 61
<210> 717
<211> 60
<212> DNA
<213> Homo sapiens
<400> 717
ctccctcccg cgctccccgc gctcgggcgc cgcagagctg tccagcttca gtgccgaacc 60
<210> 718
<211> 60
<212> DNA
<213> Homo sapiens
<400> 718
gtactggtgt acaaggacaa ggtgactttt tttcttttcc cagattgaaa gggccaaaga 60
<210> 719
<211> 60
<212> DNA
<213> Homo sapiens
<400> 719
cctccgccgc tcagccccgg actccttacg tcagggtagc ggggtccccc ctccgcgcgg 60
<210> 720
<211> 60
<212> DNA
<213> Homo sapiens
<400> 720
ccaggagagc tcggcaagta tataaggaca gaggagcgcg ggaccaagcg gcggcgaagg 60
<210> 721
<211> 60
<212> DNA
<213> Homo sapiens
<400> 721
ttccttcagc tgtgtcttaa agtaaatctt gttgtggagc ggagccctca gctgagggag 60
<210> 722
<211> 60
<212> DNA
<213> Homo sapiens
<400> 722
gtaagtatct tcttcttccc ctcgtgagtc cctccccttt tccagaatca cttgcactgt 60
<210> 723
<211> 60
<212> DNA
<213> Homo sapiens
<400> 723
ggggcggagc ggagacagta ccttcggaga taatcctttc tcctgccgca gtggagagga 60
<210> 724
<211> 60
<212> DNA
<213> Homo sapiens
<400> 724
ccctgcctag tctccatata aaagcggcgc cgcctccccg ccctctctca ctccccgctc 60
<210> 725
<211> 60
<212> DNA
<213> Homo sapiens
<400> 725
gggcggccca gccccaggtt acgtcgtccc cagaaagaat ctggccaaca gtctggccgt 60
<210> 726
<211> 60
<212> DNA
<213> Homo sapiens
<400> 726
atgctaatac accttaattt tacgattttt tcacttttcc tccccacagc gtgagtgcat 60
<210> 727
<211> 60
<212> DNA
<213> Homo sapiens
<400> 727
taaccccagt cccctttctt ctccttccgc ccctccccaa ccccgcccca taatggatgc 60
<210> 728
<211> 527
<212> DNA
<213> Homo sapiens
<400> 728
ccccagtgga aagacgcgca ggcaaaacgc accacgtgac ggagcgtgac cgcgcgccga 60
gcccaaggtc gggcaggaag agggcctatt tcccatgatt ccttcatatt tgcatatacg 120
atacaaggct gttagagaga taattagaat taatttgact gtaaacacaa agatattagt 180
acaaaatacg tgacgtagaa agtaataatt tcttgggtag tttgcagttt taaaattatg 240
ttttaaaatg gactatcata tgcttaccgt aacttgaaag tatttcgatt tcttggcttt 300
atatatcttg tggaaaggac gaaacaccgt gctcgcttcg gcagcacata tactaaaatt 360
ggaacgatac agagaagatt agcatggccc ctgcgcaagg atgacacgca aattcgtgaa 420
gcgttccata tttttacatc aggttgtttt tctgttttta catcaggttg tttttctgtt 480
tggttttttt tttacaccac gtttatacgc cggtgcacgg tttacca 527
<210> 729
<211> 250
<212> DNA
<213> Homo sapiens
<400> 729
gagggcctat ttcccatgat tccttcatat ttgcatatac gatacaaggc tgttagagag 60
ataattagaa ttaatttgac tgtaaacaca aagatattag tacaaaatac gtgacgtaga 120
aagtaataat ttcttgggta gtttgcagtt ttaaaattat gttttaaaat ggactatcat 180
atgcttaccg taacttgaaa gtatttcgat ttcttggctt tatatatctt gtggaaagga 240
cgaaacaccg 250
<210> 730
<211> 112
<212> DNA
<213> Homo sapiens
<400> 730
gagggcctat ttcccatgat tccttcatat ttgcatatac gatagcttac cgtaacttga 60
aagtatttcg atttcttggc tttatatatc ttgtggaaag gacgaaacac cg 112
<210> 731
<211> 47
<212> DNA
<213> Homo sapiens
<400> 731
tttcgatttc ttggctttat atatcttgtg gaaaggacga aacaccg 47
<210> 732
<211> 76
<212> DNA
<213> Homo sapiens
<400> 732
atacgatagc ttaccgtaac ttgaaagtat ttcgatttct tggctttata tatcttgtgg 60
aaaggacgaa acaccg 76
<210> 733
<211> 80
<212> DNA
<213> Homo sapiens
<400> 733
gagggcctat ttcccatgat tccttcatat ttgcattttc gatttcttgg ctttatatat 60
cttgtggaaa ggacgaaacg 80
<210> 734
<211> 100
<212> DNA
<213> Homo sapiens
<400> 734
aatatttgca tgtcgctatg tgttctggga aatcaccata aacgtgaaat gtctttggat 60
ttgggaatct tataagttct gtatgagacc actctttccc 100
<210> 735
<211> 244
<212> DNA
<213> Homo sapiens
<400> 735
ctgcagtatt tagcatgccc cacccatctg caagtgcatt ctggatagtg tcaaaacagg 60
cggaaatcaa gtccgtttat ctcaaacttt agcattttgg gaataaatga tatttgctat 120
gctggttaaa ttagatttta gttaaatttc ctgatgaagc tctagtacga taagcaactt 180
gacctaagtg taaagttgag atttccttca ggtttatata gcttgtgcgc cgcctgggta 240
cctc 244
<210> 736
<211> 723
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 736
aggctcagag gcacacagga gtttctgggc tcaccctgcc cccttccaac ccctcagttc 60
ccatcctcca gcagctgttt gtgtgctgcc tctgaagtcc acactgaaca aacttcagcc 120
tactcatgtc cctaaaatgg gcaaacattg caagcagcaa acagcaaaca cacagccctc 180
cctgcctgct gaccttggag ctggggcaga ggtcagagac ctctctgggc ccatgccacc 240
tccaacatcc actcgacccc ttggaatttc ggtggagagg agcagaggtt gtcctggcgt 300
ggtttaggta gtgtgagagg ggtacccggg gatcttgcta ccagtggaac agccactaag 360
gattctgcag tgagagcaga gggccagcta agtggtactc tcccagagac tgtctgactc 420
acgccacccc ctccaccttg gacacaggac gctgtggttt ctgagccagg tacaatgact 480
cctttcggta agtgcagtgg aagctgtaca ctgcccaggc aaagcgtccg ggcagcgtag 540
gcgggcgact cagatcccag ccagtggact tagcccctgt ttgctcctcc gataactggg 600
gtgaccttgg ttaatattca ccagcagcct cccccgttgc ccctctggat ccactgctta 660
aatacggacg aggacagggc cctgtctcct cagcttcagg caccaccact gacctgggac 720
agt 723
<210> 737
<211> 1725
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 737
gacattgatt attgactagt tattaatagt aatcaattac ggggtcatta gttcatagcc 60
catatatgga gttccgcgtt acataactta cggtaaatgg cccgcctggc tgaccgccca 120
acgacccccg cccattgacg tcaataatga cgtatgttcc catagtaacg ccaataggga 180
ctttccattg acgtcaatgg gtggagtatt tacggtaaac tgcccacttg gcagtacatc 240
aagtgtatca tatgccaagt acgcccccta ttgacgtcaa tgacggtaaa tggcccgcct 300
ggcattatgc ccagtacatg accttatggg actttcctac ttggcagtac atctacgtat 360
tagtcatcgc tattaccatg gtcgaggtga gccccacgtt ctgcttcact ctccccatct 420
cccccccctc cccaccccca attttgtatt tatttatttt ttaattattt tgtgcagcga 480
tgggggcggg gggggggggg gggcgcgcgc caggcggggc ggggcggggc gaggggcggg 540
gcggggcgag gcggagaggt gcggcggcag ccaatcagag cggcgcgctc cgaaagtttc 600
cttttatggc gaggcggcgg cggcggcggc cctataaaaa gcgaagcgcg cggcgggcgg 660
gagtcgctgc gcgctgcctt cgccccgtgc cccgctccgc cgccgcctcg cgccgcccgc 720
cccggctctg actgaccgcg ttactcccac aggtgagcgg gcgggacggc ccttctcctc 780
cgggctgtaa ttagcgcttg gtttaatgac ggcttgtttc ttttctgtgg ctgcgtgaaa 840
gccttgaggg gctccgggag ggccctttgt gcggggggag cggctcgggg ggtgcgtgcg 900
tgtgtgtgtg cgtggggagc gccgcgtgcg gctccgcgct gcccggcggc tgtgagcgct 960
gcgggcgcgg cgcggggctt tgtgcgctcc gcagtgtgcg cgaggggagc gcggccgggg 1020
gcggtgcccc gcggtgcggg gggggctgcg aggggaacaa aggctgcgtg cggggtgtgt 1080
gcgtgggggg gtgagcaggg ggtgtgggcg cgtcggtcgg gctgcaaccc cccctgcacc 1140
cccctccccg agttgctgag cacggcccgg cttcgggtgc ggggctccgt acggggcgtg 1200
gcgcggggct cgccgtgccg ggcggggggt ggcggcaggt gggggtgccg ggcggggcgg 1260
ggccgcctcg ggccggggag ggctcggggg aggggcgcgg cggcccccgg agcgccggcg 1320
gctgtcgagg cgcggcgagc cgcagccatt gccttttatg gtaatcgtgc gagagggcgc 1380
agggacttcc tttgtcccaa atctgtgcgg agccgaaatc tgggaggcgc cgccgcaccc 1440
cctctagcgg gcgcggggcg aagcggtgcg gcgccggcag gaaggaaatg ggcggggagg 1500
gccttcgtgc gtcgccgcgc cgccgtcccc ttctccctct ccagcctcgg ggctgtccgc 1560
ggggggacgg ctgccttcgg gggggacggg gcagggcggg gttcggcttc tggcgtgtga 1620
ccggcggctc tagagcctct gctaaccatg ttcatgcctt cttctttttc ctacagctcc 1680
tgggcaacgt gctggttatt gtgctgtctc atcattttgg caaag 1725
<210> 738
<211> 594
<212> DNA
<213> Gallus gallus
<400> 738
cgcgtggtac ctctggtcgt tacataactt acggtaaatg gcccgcctgg ctgaccgccc 60
aacgaccccg cccattgacg tcaataatga cgtatgttcc catagtaacg ccaataggga 120
ctttccattg acgtcaatgg gtggagtatt tacggtaaac tgcccacttg gcagtacatc 180
aagtgtatca tatgccaagt acgcccccta ttgacgtcaa tgacggtaaa tggcccgcct 240
ggcattatgc ccagtacatg accttatggg actttcctac ttggcagtac atctactcga 300
ggccacgttc tgcttcactc tccccatctc ccccccctcc ccacccccaa ttttgtattt 360
atttattttt taattatttt gtgcagcgat gggggcgggg gggggggggg ggggggcgcg 420
cgccaggcgg ggcggggcgg ggcgaggggc ggggcggggc gaggcggaga ggtgcggcgg 480
cagccaatca gagcggcgcg ctccgaaagt ttccttttat ggcgaggcgg cggcggcggc 540
ggccctataa aaagcgaagc gcgcggcggg cgggagcggg atcagccacc gcgg 594
<210> 739
<211> 865
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 739
ccactacggg tttaggctgc ccatgtaagg aggcaaggcc tggggacacc cgagatgcct 60
ggttataatt aacccagaca tgtggctgcc cccccccccc ccaacacctg ctgcctctaa 120
aaataaccct gtccctggtg gatcccacta cgggtttagg ctgcccatgt aaggaggcaa 180
ggcctgggga cacccgagat gcctggttat aattaaccca gacatgtggc tgcccccccc 240
ccccccaaca cctgctgcct ctaaaaataa ccctgtccct ggtggatccc actacgggtt 300
taggctgccc atgtaaggag gcaaggcctg gggacacccg agatgcctgg ttataattaa 360
cccagacatg tggctgcccc cccccccccc aacacctgct gcctctaaaa ataaccctgt 420
ccctggtgga tcccctgcat gcgaagatct tcgaacaagg ctgtggggga ctgagggcag 480
gctgtaacag gcttgggggc cagggcttat acgtgcctgg gactcccaaa gtattactgt 540
tccatgttcc cggcgaaggg ccagctgtcc cccgccagct agactcagca cttagtttag 600
gaaccagtga gcaagtcagc ccttggggca gcccatacaa ggccatgggg ctgggcaagc 660
tgcacgcctg ggtccggggt gggcacggtg cccgggcaac gagctgaaag ctcatctgct 720
ctcaggggcc cctccctggg gacagcccct cctggctagt cacaccctgt aggctcctct 780
atataaccca ggggcacagg ggctgccctc attctaccac cacctccaca gcacagacag 840
acactcagga gccagccagc gtcga 865
<210> 740
<211> 251
<212> DNA
<213> Mus musculus
<400> 740
atggaggcgg tactatgtag atgagaattc aggagcaaac tgggaaaagc aactgcttcc 60
aaatatttgt gatttttaca gtgtagtttt ggaaaaactc ttagcctacc aattcttcta 120
agtgttttaa aatgtgggag ccagtacaca tgaagttata gagtgtttta atgaggctta 180
aatatttacc gtaactatga aatgctacgc atatcatgct gttcaggctc cgtggccacg 240
caactcatac t 251
<210> 741
<211> 212
<212> DNA
<213> Homo sapiens
<400> 741
gggcagagcg cacatcgccc acagtccccg agaagttggg gggaggggtc ggcaattgaa 60
cgggtgccta gagaaggtgg cgcggggtaa actgggaaag tgatgtcgtg tactggctcc 120
gcctttttcc cgagggtggg ggagaaccgt atataagtgc agtagtcgcc gtgaacgttc 180
tttttcgcaa cgggtttgcc gccagaacac ag 212
<210> 742
<211> 460
<212> DNA
<213> Homo sapiens
<400> 742
gggctggaag ctacctttga catcatttcc tctgcgaatg catgtataat ttctacagaa 60
cctattagaa aggatcaccc agcctctgct tttgtacaac tttcccttaa aaaactgcca 120
attccactgc tgtttggccc aatagtgaga actttttcct gctgcctctt ggtgcttttg 180
cctatggccc ctattctgcc tgctgaagac actcttgcca gcatggactt aaacccctcc 240
agctctgaca atcctctttc tcttttgttt tacatgaagg gtctggcagc caaagcaatc 300
actcaaagtt caaaccttat cattttttgc tttgttcctc ttggccttgg ttttgtacat 360
cagctttgaa aataccatcc cagggttaat gctggggtta atttataact aagagtgctc 420
tagttttgca atacaggaca tgctataaaa atggaaagat 460
<210> 743
<211> 133
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 743
gtaagtatca aggttacaag acaggtttaa ggagaccaat agaaactggg cttgtcgaga 60
cagagaagac tcttgcgttt ctgataggca cctattggtc ttactgacat ccactttgcc 120
tttctctcca cag 133
<210> 744
<211> 82
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 744
gtgagtatct cagggatcca gacatgggga tatgggaggt gcctctgatc ccagggctca 60
ctgtgggtct ctctgttcac ag 82
<210> 745
<211> 304
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 745
cgttacataa cttacggtaa atggcccgcc tggctgaccg cccaacgacc cccgcccatt 60
gacgtcaata atgacgtatg ttcccatagt aacgccaata gggactttcc attgacgtca 120
atgggtggag tatttacggt aaactgccca cttggcagta catcaagtgt atcatatgcc 180
aagtacgccc cctattgacg tcaatgacgg taaatggccc gcctggcatt atgcccagta 240
catgacctta tgggactttc ctacttggca gtacatctac gtattagtca tcgctattac 300
catg 304
<210> 746
<211> 130
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 746
ctgcgcgctc gctcgctcac tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtggccaa ctccatcact 120
aggggttcct 130
<210> 747
<211> 106
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 747
ctgcgcgctc gctcgctcac tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtgg 106
<210> 748
<211> 143
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 748
gaacccctag tgatggagtt ggccactccc tctctgcgcg ctcgctcgct cactgaggcc 60
gcccgggcaa agcccgggcg tcgggcgacc tttggtcgcc cggcctcagt gagcgagcga 120
gcgcgcagag agggagtggc caa 143
<210> 749
<211> 110
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 749
ttggccactc cctctctgcg cgctcgctcg ctcactgagg ccgggcgacc aaaggtcgcc 60
cgacgcccgg gctttgcccg ggcggcctca gtgagcgagc gagcgcgcag 110
<210> 750
<211> 55
<212> DNA
<213> Oryctolagus cuniculus
<400> 750
ataaaggaaa tttattttca ttgcaatagt gtgttggaat tttttgtgtc tctca 55
<210> 751
<211> 127
<212> DNA
<213> Oryctolagus cuniculus
<400> 751
gatctttttc cctctgccaa aaattatggg gacatcatga agccccttga gcatctgact 60
tctggctaat aaaggaaatt tattttcatt gcaatagtgt gttggaattt tttgtgtctc 120
tcactcg 127
<210> 752
<211> 128
<212> DNA
<213> Homo sapiens
<400> 752
gtttgaatga ggcttcagta ctttacagaa tcgttgcctg cacatcttgg aaacacttgc 60
tgggattact tcttcaggtt aacccaacag aaggctcgag aaggtatatt gctgttgaca 120
gtgagcgc 128
<210> 753
<211> 122
<212> DNA
<213> Homo sapiens
<400> 753
ttgcctactg cctcggaatt caaggggcta ctttaggagc aattatcttg tttactaaaa 60
ctgaatacct tgctatctct ttgatacatt tttacaaagc tgaattaaaa tggtataaat 120
ta 122
<210> 754
<211> 129
<212> DNA
<213> Homo sapiens
<400> 754
gtttgaatga ggcttcagta ctttacagaa tcgttgcctg cacatcttgg aaacacttgc 60
tgggattact tcttcaggtt aacccaacag aaggctcgag aaggtatatt gctgttgaca 120
gtgagcgac 129
<210> 755
<211> 123
<212> DNA
<213> Homo sapiens
<400> 755
gctgcctact gcctcggaat tcaaggggct actttaggag caattatctt gtttactaaa 60
actgaatacc ttgctatctc tttgatacat ttttacaaag ctgaattaaa atggtataaa 120
tta 123
<210> 756
<211> 128
<212> DNA
<213> Homo sapiens
<400> 756
gtttgaatga ggcttcagta ctttacagaa tcgttgcctg cacatcttgg aaacacttgc 60
tgggattact tcttcaggtt aacccaacag aaggctcgag aaggtatatt gctgttgaca 120
gtgagcga 128
<210> 757
<211> 121
<212> DNA
<213> Homo sapiens
<400> 757
tgcctactgc ctcggaattc aaggggctac tttaggagca attatcttgt ttactaaaac 60
tgaatacctt gctatctctt tgatacattt ttacaaagct gaattaaaat ggtataaatt 120
a 121
<210> 758
<211> 17
<212> DNA
<213> Homo sapiens
<400> 758
tagtgaagcc acagatg 17
<210> 759
<211> 129
<212> DNA
<213> Homo sapiens
<400> 759
gtttgaatga ggcttcagta ctttacagaa tcgttgcctg cacatcttgg aaacacttgc 60
tgggattact tcttcaggtt aacccaacag aaggctaaag aaggtatatt gctgttgaca 120
gtgagcgac 129
<210> 760
<211> 123
<212> DNA
<213> Homo sapiens
<400> 760
gctgcctact gcctcggact tcaaggggct actttaggag caattatctt gtttactaaa 60
actgaatacc ttgctatctc tttgatacat ttttacaaag ctgaattaaa atggtataaa 120
tta 123
<210> 761
<211> 19
<212> DNA
<213> Homo sapiens
<400> 761
ctgtgaagcc acagatggg 19
<210> 762
<211> 84
<212> DNA
<213> Homo sapiens
<400> 762
caaaccagga aggggaaatc tgtggtttaa attctttatg cctcatcctc tgagtgctga 60
aggcttgctg taggctgtat gctg 84
<210> 763
<211> 203
<212> DNA
<213> Homo sapiens
<400> 763
cagtgtatga tgcctgttac tagcattcac atggaacaaa ttgctgccgt gggaggatga 60
caaagaagca tgagtcaccc tgctggataa acttagactt caggctttat catttttcaa 120
tctgttaatc ataatctggt cactgggatg ttcaacctta aactaagttt tgaaagtaag 180
gttatttaaa agatttatca gta 203
<210> 764
<211> 13
<212> DNA
<213> Homo sapiens
<400> 764
tttgcctcca act 13
<210> 765
<211> 106
<212> DNA
<213> Homo sapiens
<400> 765
acgtttccag aacgtctgta gcttttctcc tccttccctc cattttcctc ttggtcttac 60
ctttggccta gtggttggtg tagtgataat gtagcgagat tttctg 106
<210> 766
<211> 124
<212> DNA
<213> Homo sapiens
<400> 766
tggaacgtca cgcagctttc tacagcatga caagctgctg aggcttaaat caggattttc 60
ctgtctcttt ctacaaaatc aaaatgaaaa aagagggctt tttaggcatc tccgagatta 120
tgtg 124
<210> 767
<211> 20
<212> DNA
<213> Homo sapiens
<400> 767
ggttgcgagg tatgagtaaa 20
<210> 768
<211> 212
<212> DNA
<213> Homo sapiens
<400> 768
tctgccgcgg aaaggggaga agtgtgggct cctccgagtc gggggcggac tgggacagca 60
cagtcggctg agcgcagcgc ccccgccctg cccgccacgc ggcgaagacg cctgagcgtt 120
cgcgcccctc gggcgaggac cccacgcaag cccgagccgg tcccgaccct ggccccgacg 180
ctcgccgccc gccccagccc tgagggcccc tc 212
<210> 769
<211> 213
<212> DNA
<213> Homo sapiens
<400> 769
gagaggcgcc tccgccgctc ctttctcatg gaaatggccc gcgagcccgt ccggcccagc 60
gcccctcccg cgggaggaag gcgagcccgg cccccggcgg ccattcgcgc cgcggacaaa 120
tccggcgaac aatgcgcccg cccagagtgc ggcccagctg ccgggccggg gatctggccg 180
cgggacacaa aggggcccgc acgcctctgg cgt 213
<210> 770
<211> 18
<212> DNA
<213> Homo sapiens
<400> 770
atttaatgtc tatacaat 18
<210> 771
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 771
tttgtgaggg tctggtc 17
<210> 772
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 772
aaarcaggca ttagctatg 19
<210> 773
<211> 19
<212> DNA
<213> Homo sapiens
<400> 773
aaaacaggca ttagctatg 19
<210> 774
<211> 19
<212> DNA
<213> Mus musculus
<400> 774
aaagcaggca ttagctatg 19
<210> 775
<211> 1541
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 775
ctgcgcgctc gctcgctcac tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtggccaa ctccatcact 120
aggggttcct tgtagttaat gattaacccg ccatgctact tatctaccag ggtaatgggg 180
atcctctaga tccggtcggg cccgcggtac cgtcgagaag cttgatgtgg gcggagcttc 240
gaaggggcgg gcgcccgtgg ggcgggtcct gagtgggggc gggaccgggg ccggcacctg 300
ggtgaggttc tgcagagggc cctgcgtatg agtgcaagtg ggttttagga ccaggatgag 360
gcggggtggg ggtgcctacc tgacgaccga ccccgaccca ctggacaagc acccaacccc 420
cattccccaa attgcgcatc ccctatcaga gagggggagg ggaaacagga tgcggcgagg 480
cgcgtgcgca ctgccagctt cagcaccgcg gacagtgcct tcgcccccgc ctggcggcgc 540
gcgccaccgc cgcctcagca ctgaaggcgc gctgacgtca ctcgccggtc ccccgcaaac 600
tccccttccc ggccaccttg gtcgcgtccg cgccgccgcc ggcccagccg gaccgcacca 660
cgcgaggcgc gagatagggg ggcacgggcg cgaccatctg cgctgcggcg ccggcgactc 720
agcgctgcct cagtctgcca attgcagcgg aggagtcgtg tcgtgcctga gagcgcaggg 780
cgcgcctagc ccgggctagg tcgactcgac tagggataac agggtaattg tttgaatgag 840
gcttcagtac tttacagaat cgttgcctgc acatcttgga aacacttgct gggattactt 900
cttcaggtta acccaacaga aggctcgaga aggtatattg ctgttgacag tgagcgctaa 960
aacaggcatt agctatgggt agtgaagcca cagatgccca tagctaatgc ctgttttatt 1020
gcctactgcc tcggaattca aggggctact ttaggagcaa ttatcttgtt tactaaaact 1080
gaataccttg ctatctcttt gatacatttt tacaaagctg aattaaaatg gtataaatta 1140
tcacgggatc cgatcttttt ccctctgcca aaaattatgg ggacatcatg aagccccttg 1200
agcatctgac ttctggctaa taaaggaaat ttattttcat tgcaatagtg tgttggaatt 1260
ttttgtgtct ctcactcggc ggccgcccga gtttaattgg tttatagaac tcttcaagct 1320
agcgaagcaa ttcgttgatc tgaatttcga ccacccataa tacccattac cctggtagat 1380
aagtagcatg gcgggttaat cattaactac aaggaacccc tagtgatgga gttggccact 1440
ccctctctgc gcgctcgctc gctcactgag gccgggcgac caaaggtcgc ccgacgcccg 1500
ggctttgccc gggcggcctc agtgagcgag cgagcgcgca g 1541
<210> 776
<400> 776
000
<210> 777
<211> 2893
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 777
ctgcgcgctc gctcgctcac tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtggccaa ctccatcact 120
aggggttcct tgtagttaat gattaacccg ccatgctact tatctaccag ggtaatgggg 180
atcctctaga tccggtcggg cccgcggtac cgtcgagaag cttgatgtgg gcggagcttc 240
gaaggggcgg gcgcccgtgg ggcgggtcct gagtgggggc gggaccgggg ccggcacctg 300
ggtgaggttc gatcctatca cgagactagc ctcgagaagc ttgatatcag cacccacata 360
gcagctcaca aatgtctgaa actccaattc ttgggaatct gacacgatca cacatgcagg 420
caaaatacca atgtacatga attaaaaaaa aaaaaaacaa cctttaaaag aaacaagggt 480
tcagtaccac tactgacatc ttgtttcccc agaggcctta ctttaattat ttattgtttc 540
cacttagttg ctcaattaat taatttagag gtttttttct tcctttcttt ttcttttttc 600
tttctctctt ttttttcttc ttaagacagg gtttctctgt gtagctcagg ctatcctgga 660
actcactctg tagaccaggc tggccttgta ctcaaagatc tgcctgcctc tgcctcccca 720
gtgctgggat taaagacatg caccatcact gccctgcttt cctcttttta ttttgaaaat 780
tgttcatcaa cagttactaa acgtgttcga attccaagag ctgactagac atataagacc 840
attcagcctt ctgaataaga tgtaggtgtg cccctcctct tactcctcta tttggaagtt 900
ggttactttc tgtatgtagt atgcgaatcc ccctctgcca ccccgctttc tgttttaaaa 960
cagaaaaggc tgcaacatac agtgtgtgct tctgttcttg aactggaagc ttaggctgtc 1020
ctggacttgg gttgagacct gggctcatcc agataggaaa tggatttggt gaccccgcca 1080
ggacttcgca ggcaccacat cgtggtcgtg tgtgggtgct gtatgcaccc actgattgcg 1140
cgcgtgggtt ccagagcttg gtggtctgcg agaggagagt gggcaagagt gggtgtgtct 1200
gtggagcccc agctaggggc tgctgcccgc tgctcccact tgtggctcct gggcgccgcc 1260
agcaggcaca tctccggagg acgccgcggg atgggagctg atgacaggag agcgccgtct 1320
cccgagtgat ggcagcgcac gctgctgcct cgccgcctcc gccgctcagt cctgatctta 1380
cgttagggta gctgggtacc ccctccgccc gggaaccagc tagtagaggg agaacagagc 1440
agagcgtgcg gcagagccga tcccgcgtcc cgccgaaccc tgccaagccc cgccaatccc 1500
agcagagcag gaaccagcgc agctgagcca acaccggacg ccgcactgag acccagcatt 1560
ccccagccgc cactacccgg tccccgccgg ggtgccgggc tcgtcctgtg agcccctcgt 1620
catgcgtgtc gggctcttcg actctccaga tcagttccag agcgctgagg gccctgcgta 1680
tgagtgcaag tgggttttag gaccaggatg aggcggggtg ggggtgccta cctgacgacc 1740
gaccccgacc cactggacaa gcacccaacc cccattcccc aaattgcgca tcccctatca 1800
gagaggggga ggggaaacag gatgcggcga ggcgcgtgcg cactgccagc ttcagcaccg 1860
cggacagtgc cttcgccccc gcctggcggc gcgcgccacc gccgcctcag cactgaaggc 1920
gcgctgacgt cactcgccgg tcccccgcaa actccccttc ccggccacct tggtcgcgtc 1980
cgcgccgccg ccggcccagc cggaccgcac cacgcgaggc gcgagatagg ggggcacggg 2040
cgcgaccatc tgcgctgcgg cgccggcgac tcagcgctgc ctcagtctgc caattgcagc 2100
ggaggagtcg tgtcgtgcct gagagcgcag ggcgcgccta gcccgggcta ggtcgactcg 2160
actagggata acagggtaat tgtttgaatg aggcttcagt actttacaga atcgttgcct 2220
gcacatcttg gaaacacttg ctgggattac ttcttcaggt taacccaaca gaaggctcga 2280
gaaggtatat tgctgttgac agtgagcgct aaaacaggca ttagctatgg gtagtgaagc 2340
cacagatgcc catagctaat gcctgtttta ttgcctactg cctcggaatt caaggggcta 2400
ctttaggagc aattatcttg tttactaaaa ctgaatacct tgctatctct ttgatacatt 2460
tttacaaagc tgaattaaaa tggtataaat tatcacggga tccgatcttt ttccctctgc 2520
caaaaattat ggggacatca tgaagcccct tgagcatctg acttctggct aataaaggaa 2580
atttattttc attgcaatag tgtgttggaa ttttttgtgt ctctcactcg gcggccgccc 2640
gagtttaatt ggtttataga actcttcaag ctagcgaagc aattcgttga tctgaatttc 2700
gaccacccat aatacccatt accctggtag ataagtagca tggcgggtta atcattaact 2760
acaaggaacc cctagtgatg gagttggcca ctccctctct gcgcgctcgc tcgctcactg 2820
aggccgggcg accaaaggtc gcccgacgcc cgggctttgc ccgggcggcc tcagtgagcg 2880
agcgagcgcg cag 2893
<210> 778
<211> 311
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 778
gtttgaatga ggcttcagta ctttacagaa tcgttgcctg cacatcttgg aaacacttgc 60
tgggattact tcttcaggtt aacccaacag aaggctcgag aaggtatatt gctgttgaca 120
gtgagcgcta aaacaggcat tagctatggg tagtgaagcc acagatgccc atagctaatg 180
cctgttttat tgcctactgc ctcggaattc aaggggctac tttaggagca attatcttgt 240
ttactaaaac tgaatacctt gctatctctt tgatacattt ttacaaagct gaattaaaat 300
ggtataaatt a 311
<210> 779
<211> 1289
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 779
ctgcgcgctc gctcgctcac tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtggaatt cacgcgtggt 120
accctgcaga gggccctgcg tatgagtgca agtgggtttt aggaccagga tgaggcgggg 180
tgggggtgcc tacctgacga ccgaccccga cccactggac aagcacccaa cccccattcc 240
ccaaattgcg catcccctat cagagagggg gaggggaaac aggatgcggc gaggcgcgtg 300
cgcactgcca gcttcagcac cgcggacagt gccttcgccc ccgcctggcg gcgcgcgcca 360
ccgccgcctc agcactgaag gcgcgctgac gtcactcgcc ggtcccccgc aaactcccct 420
tcccggccac cttggtcgcg tccgcgccgc cgccggccca gccggaccgc accacgcgag 480
gcgcgagata ggggggcacg ggcgcgacca tctgcgctgc ggcgccggcg actcagcgct 540
gcctcagtct gccaattgca gcggaggagt cgtgtcgtgc ctgagagcgc agggcgcgcc 600
tagcccgggc taggtcgact cgactaggga taacagggta attgtttgaa tgaggcttca 660
gtactttaca gaatcgttgc ctgcacatct tggaaacact tgctgggatt acttcttcag 720
gttaacccaa cagaaggctc gagaaggtat attgctgttg acagtgagcg ctaaaacagg 780
cattagctat gggtagtgaa gccacagatg cccatagcta atgcctgttt tattgcctac 840
tgcctcggaa ttcaaggggc tactttagga gcaattatct tgtttactaa aactgaatac 900
cttgctatct ctttgataca tttttacaaa gctgaattaa aatggtataa attatcacgg 960
gatccaagct tgatcttttt ccctctgcca aaaattatgg ggacatcatg aagccccttg 1020
agcatctgac ttctggctaa taaaggaaat ttattttcat tgcaatagtg tgttggaatt 1080
ttttgtgtct ctcactcggc tagcgaagca attctagcag gcatgctggg gagagatcga 1140
tctgaggaac ccctagtgat ggagttggcc actccctctc tgcgcgctcg ctcgctcact 1200
gaggccgccc gggcaaagcc cgggcgtcgg gcgacctttg gtcgcccggc ctcagtgagc 1260
gagcgagcgc gcagagaggg agtggccaa 1289
<210> 780
<211> 311
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 780
gtttgaatga ggcttcagta ctttacagaa tcgttgcctg cacatcttgg aaacacttgc 60
tgggattact tcttcaggtt aacccaacag aaggctcgag aaggtatatt gctgttgaca 120
gtgagcgcta aaacaggcat tagctatggg tagtgaagcc acagatgccc atagctaatg 180
cctgttttat tgcctactgc ctcggaattc aaggggctac tttaggagca attatcttgt 240
ttactaaaac tgaatacctt gctatctctt tgatacattt ttacaaagct gaattaaaat 300
ggtataaatt a 311
<210> 781
<211> 1290
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 781
ctgcgcgctc gctcgctcac tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtggaatt cacgcgtggt 120
acccggccgc cgagtgagag acacaaaaaa ttccaacaca ctattgcaat gaaaataaat 180
ttcctttatt agccagaagt cagatgctca aggggcttca tgatgtcccc ataatttttg 240
gcagagggaa aaagatcgga tcccgtgata atttatacca ttttaattca gctttgtaaa 300
aatgtatcaa agagatagca aggtattcag ttttagtaaa caagataatt gctcctaaag 360
tagccccttg aattccgagg cagtaggcaa taaaacaggc attagctatg ggcatctgtg 420
gcttcactac ccatagctaa tgcctgtttt agcgctcact gtcaacagca atataccttc 480
tcgagccttc tgttgggtta acctgaagaa gtaatcccag caagtgtttc caagatgtgc 540
aggcaacgat tctgtaaagt actgaagcct cattcaaaca attaccctgt tatccctagt 600
cgagtcgacc tagcccgggc taggcgcgcc ctgcgctctc aggcacgaca cgactcctcc 660
gctgcaattg gcagactgag gcagcgctga gtcgccggcg ccgcagcgca gatggtcgcg 720
cccgtgcccc cctatctcgc gcctcgcgtg gtgcggtccg gctgggccgg cggcggcgcg 780
gacgcgacca aggtggccgg gaaggggagt ttgcggggga ccggcgagtg acgtcagcgc 840
gccttcagtg ctgaggcggc ggtggcgcgc gccgccaggc gggggcgaag gcactgtccg 900
cggtgctgaa gctggcagtg cgcacgcgcc tcgccgcatc ctgtttcccc tccccctctc 960
tgatagggga tgcgcaattt ggggaatggg ggttgggtgc ttgtccagtg ggtcggggtc 1020
ggtcgtcagg taggcacccc caccccgcct catcctggtc ctaaaaccca cttgcactca 1080
tacgcagggc cctctgcagg ctagcgaagc aattctagca ggcatgctgg ggagagatcg 1140
atctgaggaa cccctagtga tggagttggc cactccctct ctgcgcgctc gctcgctcac 1200
tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt ggtcgcccgg cctcagtgag 1260
cgagcgagcg cgcagagagg gagtggccaa 1290
<210> 782
<211> 311
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 782
taatttatac cattttaatt cagctttgta aaaatgtatc aaagagatag caaggtattc 60
agttttagta aacaagataa ttgctcctaa agtagcccct tgaattccga ggcagtaggc 120
aataaaacag gcattagcta tgggcatctg tggcttcact acccatagct aatgcctgtt 180
ttagcgctca ctgtcaacag caatatacct tctcgagcct tctgttgggt taacctgaag 240
aagtaatccc agcaagtgtt tccaagatgt gcaggcaacg attctgtaaa gtactgaagc 300
ctcattcaaa c 311
<210> 783
<211> 2247
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 783
ctgcgcgctc gctcgctcac tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtggccaa ctccatcact 120
aggggttcct tgtagttaat gattaacccg ccatgctact tatctaccag ggtaatgggg 180
atcctctaga tccggtcggg cccgcggtac cgtcgagaag cttgatgtgg gcggagcttc 240
gaaggggcgg gcgcccgtgg ggcgggtcct gagtgggggc gggaccgggg ccggcacctg 300
ggtgaggttc tgcagagggc cctgcgtatg agtgcaagtg ggttttagga ccaggatgag 360
gcggggtggg ggtgcctacc tgacgaccga ccccgaccca ctggacaagc acccaacccc 420
cattccccaa attgcgcatc ccctatcaga gagggggagg ggaaacagga tgcggcgagg 480
cgcgtgcgca ctgccagctt cagcaccgcg gacagtgcct tcgcccccgc ctggcggcgc 540
gcgccaccgc cgcctcagca ctgaaggcgc gctgacgtca ctcgccggtc ccccgcaaac 600
tccccttccc ggccaccttg gtcgcgtccg cgccgccgcc ggcccagccg gaccgcacca 660
cgcgaggcgc gagatagggg ggcacgggcg cgaccatctg cgctgcggcg ccggcgactc 720
agcgctgcct cagtctgcca attgcagcgg aggagtcgtg tcgtgcctga gagcgcaggg 780
cgcgcctagc ccgggctagg tcgactcgac tagggataac agggtaattg tttgaatgag 840
gcttcagtac tttacagaat cgttgcctgc acatcttgga aacacttgct gggattactt 900
cttcaggtta acccaacaga aggctcgaga aggtatattg ctgttgacag tgagcgctaa 960
aacaggcatt agctatgggt agtgaagcca cagatgccca tagctaatgc ctgttttatt 1020
gcctactgcc tcggaattca aggggctact ttaggagcaa ttatcttgtt tactaaaact 1080
gaataccttg ctatctcttt gatacatttt tacaaagctg aattaaaatg gtataaatta 1140
tcacgggatc cggtcgactc gactagggat aacagggtaa ttgtttgaat gaggcttcag 1200
tactttacag aatcgttgcc tgcacatctt ggaaacactt gctgggatta cttcttcagg 1260
ttaacccaac agaaggctcg agaaggtata ttgctgttga cagtgagcgc taaaacaggc 1320
attagctatg ggtagtgaag ccacagatgc ccatagctaa tgcctgtttt attgcctact 1380
gcctcggaat tcaaggggct actttaggag caattatctt gtttactaaa actgaatacc 1440
ttgctatctc tttgatacat ttttacaaag ctgaattaaa atggtataaa ttatcacggg 1500
atccggtcga ctcgactagg gataacaggg taattgtttg aatgaggctt cagtacttta 1560
cagaatcgtt gcctgcacat cttggaaaca cttgctggga ttacttcttc aggttaaccc 1620
aacagaaggc tcgagaaggt atattgctgt tgacagtgag cgctaaaaca ggcattagct 1680
atgggtagtg aagccacaga tgcccatagc taatgcctgt tttattgcct actgcctcgg 1740
aattcaaggg gctactttag gagcaattat cttgtttact aaaactgaat accttgctat 1800
ctctttgata catttttaca aagctgaatt aaaatggtat aaattatcac gggatccgat 1860
ctttttccct ctgccaaaaa ttatggggac atcatgaagc cccttgagca tctgacttct 1920
ggctaataaa ggaaatttat tttcattgca atagtgtgtt ggaatttttt gtgtctctca 1980
ctcggcggcc gcccgagttt aattggttta tagaactctt caagctagcg aagcaattcg 2040
ttgatctgaa tttcgaccac ccataatacc cattaccctg gtagataagt agcatggcgg 2100
gttaatcatt aactacaagg aacccctagt gatggagttg gccactccct ctctgcgcgc 2160
tcgctcgctc actgaggccg ggcgaccaaa ggtcgcccga cgcccgggct ttgcccgggc 2220
ggcctcagtg agcgagcgag cgcgcag 2247
<210> 784
<211> 2247
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 784
ctgcgcgctc gctcgctcac tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtggccaa ctccatcact 120
aggggttcct tgtagttaat gattaacccg ccatgctact tatctaccag ggtaatgggg 180
atcctctaga tccggtcggg cccgcggtac cgtcgagaag cttgatgtgg gcggagcttc 240
gaaggggcgg gcgcccgtgg ggcgggtcct gagtgggggc gggaccgggg ccggcacctg 300
ggtgaggttc tgcagagggc cctgcgtatg agtgcaagtg ggttttagga ccaggatgag 360
gcggggtggg ggtgcctacc tgacgaccga ccccgaccca ctggacaagc acccaacccc 420
cattccccaa attgcgcatc ccctatcaga gagggggagg ggaaacagga tgcggcgagg 480
cgcgtgcgca ctgccagctt cagcaccgcg gacagtgcct tcgcccccgc ctggcggcgc 540
gcgccaccgc cgcctcagca ctgaaggcgc gctgacgtca ctcgccggtc ccccgcaaac 600
tccccttccc ggccaccttg gtcgcgtccg cgccgccgcc ggcccagccg gaccgcacca 660
cgcgaggcgc gagatagggg ggcacgggcg cgaccatctg cgctgcggcg ccggcgactc 720
agcgctgcct cagtctgcca attgcagcgg aggagtcgtg tcgtgcctga gagcgcaggg 780
cgcgcctagc ccgggctagg tcgactcgac tagggataac agggtaattg tttgaatgag 840
gcttcagtac tttacagaat cgttgcctgc acatcttgga aacacttgct gggattactt 900
cttcaggtta acccaacaga aggctcgaga aggtatattg ctgttgacag tgagcgctaa 960
aacaggcatt agctatgggt agtgaagcca cagatgccca tagctaatgc ctgttttatt 1020
gcctactgcc tcggaattca aggggctact ttaggagcaa ttatcttgtt tactaaaact 1080
gaataccttg ctatctcttt gatacatttt tacaaagctg aattaaaatg gtataaatta 1140
tcacgggatc cggtcgactc gactagggat aacagggtaa ttgtttgaat gaggcttcag 1200
tactttacag aatcgttgcc tgcacatctt ggaaacactt gctgggatta cttcttcagg 1260
ttaacccaac agaaggctcg agaaggtata ttgctgttga cagtgagcga catgggttct 1320
ccatatcgag actagtgaag ccacagatgg tctcgatatg gagaacccat gctgcctact 1380
gcctcggaat tcaaggggct actttaggag caattatctt gtttactaaa actgaatacc 1440
ttgctatctc tttgatacat ttttacaaag ctgaattaaa atggtataaa ttatcacggg 1500
atccggtcga ctcgactagg gataacaggg taattgtttg aatgaggctt cagtacttta 1560
cagaatcgtt gcctgcacat cttggaaaca cttgctggga ttacttcttc aggttaaccc 1620
aacagaaggc tcgagaaggt atattgctgt tgacagtgag cgaaatcctt ggctttacat 1680
atgaatagtg aagccacaga tgttcatatg taaagccaag gattctgcct actgcctcgg 1740
aattcaaggg gctactttag gagcaattat cttgtttact aaaactgaat accttgctat 1800
ctctttgata catttttaca aagctgaatt aaaatggtat aaattatcac gggatccgat 1860
ctttttccct ctgccaaaaa ttatggggac atcatgaagc cccttgagca tctgacttct 1920
ggctaataaa ggaaatttat tttcattgca atagtgtgtt ggaatttttt gtgtctctca 1980
ctcggcggcc gcccgagttt aattggttta tagaactctt caagctagcg aagcaattcg 2040
ttgatctgaa tttcgaccac ccataatacc cattaccctg gtagataagt agcatggcgg 2100
gttaatcatt aactacaagg aacccctagt gatggagttg gccactccct ctctgcgcgc 2160
tcgctcgctc actgaggccg ggcgaccaaa ggtcgcccga cgcccgggct ttgcccgggc 2220
ggcctcagtg agcgagcgag cgcgcag 2247
<210> 785
<211> 3643
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 785
ctgcgcgctc gctcgctcac tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtggccaa ctccatcact 120
aggggttcct tgtagttaat gattaacccg ccatgctact tatctaccag ggtaatgggg 180
atcctctaga tccggtcggg cccgcggtac cgtcgagaag cttgatgtgg gcggagcttc 240
gaaggggcgg gcgcccgtgg ggcgggtcct gagtgggggc gggaccgggg ccggcacctg 300
ggtgaggttc tgcagagggc cctgcgtatg agtgcaagtg ggttttagga ccaggatgag 360
gcggggtggg ggtgcctacc tgacgaccga ccccgaccca ctggacaagc acccaacccc 420
cattccccaa attgcgcatc ccctatcaga gagggggagg ggaaacagga tgcggcgagg 480
cgcgtgcgca ctgccagctt cagcaccgcg gacagtgcct tcgcccccgc ctggcggcgc 540
gcgccaccgc cgcctcagca ctgaaggcgc gctgacgtca ctcgccggtc ccccgcaaac 600
tccccttccc ggccaccttg gtcgcgtccg cgccgccgcc ggcccagccg gaccgcacca 660
cgcgaggcgc gagatagggg ggcacgggcg cgaccatctg cgctgcggcg ccggcgactc 720
agcgctgcct cagtctgcgg tgggcagcgg aggagtcgtg tcgtgcctga gagcgcaggg 780
cgcgcctgaa ttctctgccg cggaaagggg agaagtgtgg gctcctccga gtcgggggcg 840
gactgggaca gcacagtcgg ctgagcgcag cgcccccgcc ctgcccgcca cgcggcgaag 900
acgcctgagc gttcgcgccc ctcgggcgag gaccccacgc aagcccgagc cggtcccgac 960
cctggccccg acgctcgccg cccgccccag ccctgagggc ccctctacaa tgggcactag 1020
acatgggatt taatgtctat acaatcccat agctaatgcc tgttttagag aggcgcctcc 1080
gccgctcctt tctcatggaa atggcccgcg agcccgtccg gcccagcgcc cctcccgcgg 1140
gaggaaggcg agcccggccc ccggcggcca ttcgcgccgc ggacaaatcc ggcgaacaat 1200
gcgcccgccc agagtgcggc ccagctgccg ggccggggat ctggccgcgg gacacaaagg 1260
ggcccgcacg cctctggcgt ctcgagggga tccctgtgcc ttctagttgc cagccatctg 1320
ttgtttgccc ctcccccgtg ccttccttga ccctggaagg tgccactccc actgtccttt 1380
cctaataaaa tgaggaaatt gcatcgcatt gtctgagtag gtgtcattct attctggggg 1440
gtggggtggg gcaggacagc aagggggagg attgggaaga caatagcagg catgctgggg 1500
aggcggccgc ccgagtttaa ttggtttata gaactcttca agctagcact agtgaagcaa 1560
ttcgttgcat tatggcctta ggtcacttca tctccatggg gttcttcttc tgattttcta 1620
gaaaatgaga tgggggtgca gagagcttcc tcagtgacct gcccagggtc acatcagaaa 1680
tgtcagagct agaacttgaa ctcagattac taatcttaaa ttccatgcct tgggggcatg 1740
caagtacgat atacagaagg agtgaactca ttagggcaga tgaccaatga gtttaggaaa 1800
gaagagtcca gggcagggta catctacacc acccgcccag ccctgggtga gtccagccac 1860
gttcacctca ttatagttgc ctctctccag tcctaccttg acgggaagca caagcagaaa 1920
ctgggacagg agccccagga gaccaaatct tcatggtccc tctgggagga tgggtgggga 1980
gagctgtggc agaggcctca ggaggggccc tgctgctcag tggtgacaga taggggtgag 2040
aaagcagaca gagtcattcc gtcagcattc tgggtctgtt tggtacttct tctcacgcta 2100
aggtggcggt gtgatatgca caatggctaa aaagcaggga gagctggaaa gaaacaagga 2160
cagagacaga ggccaagtca accagaccaa ttcccagagg aagcaaagaa accattacag 2220
agactacaag ggggaaggga aggagagatg aattagcttc ccctgtaaac cttagaaccc 2280
agctgttgcc agggcaacgg ggcaatacct gtctcttcag aggagatgaa gttgccaggg 2340
taactacatc ctgtctttct caaggaccat cccagaatgt ggcacccact agccgttacc 2400
atagcaactg cctctttgcc ccacttaatc ccatcccgtc tgttaaaagg gccctatagt 2460
tggaggtggg ggaggtagga agagcgatga tcacttgtgg actaagtttg ttcgcatccc 2520
cttctccaac cccctcagta catcaccctg ggggaacagg gtccacttgc tcctgggccc 2580
acacagtcct gcagtattgt gtatataagg ccagggcaaa gaggagcagg ttttaaagtg 2640
aaaggcaggc aggtgttggg gaggcagtta ccggggcaac gggaacaggg cgtttcggag 2700
gtggttgcca tggggacctg gatgctgacg aaggctcgcg aggctgtgag cagccacagt 2760
gccctgctca gaagccccaa gctcgtcagt caagccggtt ctccgtttgc actcaggagc 2820
acgggcaggc gagtggcccc tagttctggg ggcagcgaat tccaattggc gcgcctagcc 2880
cgggctaggt cgactcgact agggataaca gggtaattgt ttgaatgagg cttcagtact 2940
ttacagaatc gttgcctgca catcttggaa acacttgctg ggattacttc ttcaggttaa 3000
cccaacagaa ggctaaagaa ggtatattgc tgttgacagt gagcgacgtc tcgatatgga 3060
gaacccatgc tgtgaagcca cagatgggca tgggttttat atcgagacgc tgcctactgc 3120
ctcggacttc aaggggctac tttaggagca attatcttgt ttactaaaac tgaatacctt 3180
gctatctctt tgatacattt ttacaaagct gaattaaaat ggtataaatt atcacaccgg 3240
tctcgaggga tccgatcttt ttccctctgc caaaaattat ggggacatca tgaagcccct 3300
tgagcatctg acttctggct aataaaggaa atttattttc attgcaatag tgtgttggaa 3360
ttttttgtgt ctctcactcg gcggccgccc gagtttaatt ggtttataga actcttcaag 3420
ctagcgaagc aattcgttga tctgaatttc gaccacccat aatacccatt accctggtag 3480
ataagtagca tggcgggtta atcattaact acaaggaacc cctagtgatg gagttggcca 3540
ctccctctct gcgcgctcgc tcgctcactg aggccgggcg accaaaggtc gcccgacgcc 3600
cgggctttgc ccgggcggcc tcagtgagcg agcgagcgcg cag 3643
<210> 786
<211> 3594
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 786
ctgcgcgctc gctcgctcac tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtggccaa ctccatcact 120
aggggttcct tgtagttaat gattaacccg ccatgctact tatctaccag ggtaatgggg 180
atcctctaga tccggtcggg cccgcggtac cgtcgagaag cttgatgtgg gcggagcttc 240
gaaggggcgg gcgcccgtgg ggcgggtcct gagtgggggc gggaccgggg ccggcacctg 300
ggtgaggttc tgcagagggc cctgcgtatg agtgcaagtg ggttttagga ccaggatgag 360
gcggggtggg ggtgcctacc tgacgaccga ccccgaccca ctggacaagc acccaacccc 420
cattccccaa attgcgcatc ccctatcaga gagggggagg ggaaacagga tgcggcgagg 480
cgcgtgcgca ctgccagctt cagcaccgcg gacagtgcct tcgcccccgc ctggcggcgc 540
gcgccaccgc cgcctcagca ctgaaggcgc gctgacgtca ctcgccggtc ccccgcaaac 600
tccccttccc ggccaccttg gtcgcgtccg cgccgccgcc ggcccagccg gaccgcacca 660
cgcgaggcgc gagatagggg ggcacgggcg cgaccatctg cgctgcggcg ccggcgactc 720
agcgctgcct cagtctgcgg tgggcagcgg aggagtcgtg tcgtgcctga gagcgcaggg 780
cgcgcctgaa ttctctgccg cggaaagggg agaagtgtgg gctcctccga gtcgggggcg 840
gactgggaca gcacagtcgg ctgagcgcag cgcccccgcc ctgcccgcca cgcggcgaag 900
acgcctgagc gttcgcgccc ctcgggcgag gaccccacgc aagcccgagc cggtcccgac 960
cctggccccg acgctcgccg cccgccccag ccctgagggc ccctctacaa tgggcactag 1020
acatgggatt taatgtctat acaatcccat agctaatgcc tgttttagag aggcgcctcc 1080
gccgctcctt tctcatggaa atggcccgcg agcccgtccg gcccagcgcc cctcccgcgg 1140
gaggaaggcg agcccggccc ccggcggcca ttcgcgccgc ggacaaatcc ggcgaacaat 1200
gcgcccgccc agagtgcggc ccagctgccg ggccggggat ctggccgcgg gacacaaagg 1260
ggcccgcacg cctctggcgt ctcgagggga tccctgtgcc ttctagttgc cagccatctg 1320
ttgtttgccc ctcccccgtg ccttccttga ccctggaagg tgccactccc actgtccttt 1380
cctaataaaa tgaggaaatt gcatcgcatt gtctgagtag gtgtcattct attctggggg 1440
gtggggtggg gcaggacagc aagggggagg attgggaaga caatagcagg catgctgggg 1500
aggcggccgc ccgagtttaa ttggtttata gaactcttca agctagcact agtgaagcaa 1560
ttcgttgcat tatggcctta ggtcacttca tctccatggg gttcttcttc tgattttcta 1620
gaaaatgaga tgggggtgca gagagcttcc tcagtgacct gcccagggtc acatcagaaa 1680
tgtcagagct agaacttgaa ctcagattac taatcttaaa ttccatgcct tgggggcatg 1740
caagtacgat atacagaagg agtgaactca ttagggcaga tgaccaatga gtttaggaaa 1800
gaagagtcca gggcagggta catctacacc acccgcccag ccctgggtga gtccagccac 1860
gttcacctca ttatagttgc ctctctccag tcctaccttg acgggaagca caagcagaaa 1920
ctgggacagg agccccagga gaccaaatct tcatggtccc tctgggagga tgggtgggga 1980
gagctgtggc agaggcctca ggaggggccc tgctgctcag tggtgacaga taggggtgag 2040
aaagcagaca gagtcattcc gtcagcattc tgggtctgtt tggtacttct tctcacgcta 2100
aggtggcggt gtgatatgca caatggctaa aaagcaggga gagctggaaa gaaacaagga 2160
cagagacaga ggccaagtca accagaccaa ttcccagagg aagcaaagaa accattacag 2220
agactacaag ggggaaggga aggagagatg aattagcttc ccctgtaaac cttagaaccc 2280
agctgttgcc agggcaacgg ggcaatacct gtctcttcag aggagatgaa gttgccaggg 2340
taactacatc ctgtctttct caaggaccat cccagaatgt ggcacccact agccgttacc 2400
atagcaactg cctctttgcc ccacttaatc ccatcccgtc tgttaaaagg gccctatagt 2460
tggaggtggg ggaggtagga agagcgatga tcacttgtgg actaagtttg ttcgcatccc 2520
cttctccaac cccctcagta catcaccctg ggggaacagg gtccacttgc tcctgggccc 2580
acacagtcct gcagtattgt gtatataagg ccagggcaaa gaggagcagg ttttaaagtg 2640
aaaggcaggc aggtgttggg gaggcagtta ccggggcaac gggaacaggg cgtttcggag 2700
gtggttgcca tggggacctg gatgctgacg aaggctcgcg aggctgtgag cagccacagt 2760
gccctgctca gaagccccaa gctcgtcagt caagccggtt ctccgtttgc actcaggagc 2820
acgggcaggc gagtggcccc tagttctggg ggcagcgaat tccaattggc gcgcctagcc 2880
cgggctaggt cgacacgttt ccagaacgtc tgtagctttt ctcctccttc cctccatttt 2940
cctcttggtc ttacctttgg cctagtggtt ggtgtagtga taatgtagcg agattttctg 3000
cagagcattg cagatggact gggttgcgag gtatgagtaa acagtccata cgcaatgctc 3060
cgtggaacgt cacgcagctt tctacagcat gacaagctgc tgaggcttaa atcaggattt 3120
tcctgtctct ttctacaaaa tcaaaatgaa aaaagagggc tttttaggca tctccgagat 3180
tatgtgaccg gtctcgaggg atccgatctt tttccctctg ccaaaaatta tggggacatc 3240
atgaagcccc ttgagcatct gacttctggc taataaagga aatttatttt cattgcaata 3300
gtgtgttgga attttttgtg tctctcactc ggcggccgcc cgagtttaat tggtttatag 3360
aactcttcaa gctagcgaag caattcgttg atctgaattt cgaccaccca taatacccat 3420
taccctggta gataagtagc atggcgggtt aatcattaac tacaaggaac ccctagtgat 3480
ggagttggcc actccctctc tgcgcgctcg ctcgctcact gaggccgggc gaccaaaggt 3540
cgcccgacgc ccgggctttg cccgggcggc ctcagtgagc gagcgagcgc gcag 3594
<210> 787
<211> 3649
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 787
ctgcgcgctc gctcgctcac tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtggccaa ctccatcact 120
aggggttcct tgtagttaat gattaacccg ccatgctact tatctaccag ggtaatgggg 180
atcctctaga tccggtcggg cccgcggtac cgtcgagaag cttgatgtgg gcggagcttc 240
gaaggggcgg gcgcccgtgg ggcgggtcct gagtgggggc gggaccgggg ccggcacctg 300
ggtgaggttc tgcagagggc cctgcgtatg agtgcaagtg ggttttagga ccaggatgag 360
gcggggtggg ggtgcctacc tgacgaccga ccccgaccca ctggacaagc acccaacccc 420
cattccccaa attgcgcatc ccctatcaga gagggggagg ggaaacagga tgcggcgagg 480
cgcgtgcgca ctgccagctt cagcaccgcg gacagtgcct tcgcccccgc ctggcggcgc 540
gcgccaccgc cgcctcagca ctgaaggcgc gctgacgtca ctcgccggtc ccccgcaaac 600
tccccttccc ggccaccttg gtcgcgtccg cgccgccgcc ggcccagccg gaccgcacca 660
cgcgaggcgc gagatagggg ggcacgggcg cgaccatctg cgctgcggcg ccggcgactc 720
agcgctgcct cagtctgcgg tgggcagcgg aggagtcgtg tcgtgcctga gagcgcaggg 780
cgcgcctagc ccgggctagg tcgactcgac tagggataac agggtaattg tttgaatgag 840
gcttcagtac tttacagaat cgttgcctgc acatcttgga aacacttgct gggattactt 900
cttcaggtta acccaacaga aggctaaaga aggtatattg ctgttgacag tgagcgacgt 960
ctcgatatgg agaacccatg ctgtgaagcc acagatgggc atgggtttta tatcgagacg 1020
ctgcctactg cctcggactt caaggggcta ctttaggagc aattatcttg tttactaaaa 1080
ctgaatacct tgctatctct ttgatacatt tttacaaagc tgaattaaaa tggtataaat 1140
tatcacggga tccctgtgcc ttctagttgc cagccatctg ttgtttgccc ctcccccgtg 1200
ccttccttga ccctggaagg tgccactccc actgtccttt cctaataaaa tgaggaaatt 1260
gcatcgcatt gtctgagtag gtgtcattct attctggggg gtggggtggg gcaggacagc 1320
aagggggagg attgggaaga caatagcagg catgctgggg aggcggccgc ccgagtttaa 1380
ttggtttata gaactcttca agctagcact agtgaagcaa ttcgttgcat tatggcctta 1440
ggtcacttca tctccatggg gttcttcttc tgattttcta gaaaatgaga tgggggtgca 1500
gagagcttcc tcagtgacct gcccagggtc acatcagaaa tgtcagagct agaacttgaa 1560
ctcagattac taatcttaaa ttccatgcct tgggggcatg caagtacgat atacagaagg 1620
agtgaactca ttagggcaga tgaccaatga gtttaggaaa gaagagtcca gggcagggta 1680
catctacacc acccgcccag ccctgggtga gtccagccac gttcacctca ttatagttgc 1740
ctctctccag tcctaccttg acgggaagca caagcagaaa ctgggacagg agccccagga 1800
gaccaaatct tcatggtccc tctgggagga tgggtgggga gagctgtggc agaggcctca 1860
ggaggggccc tgctgctcag tggtgacaga taggggtgag aaagcagaca gagtcattcc 1920
gtcagcattc tgggtctgtt tggtacttct tctcacgcta aggtggcggt gtgatatgca 1980
caatggctaa aaagcaggga gagctggaaa gaaacaagga cagagacaga ggccaagtca 2040
accagaccaa ttcccagagg aagcaaagaa accattacag agactacaag ggggaaggga 2100
aggagagatg aattagcttc ccctgtaaac cttagaaccc agctgttgcc agggcaacgg 2160
ggcaatacct gtctcttcag aggagatgaa gttgccaggg taactacatc ctgtctttct 2220
caaggaccat cccagaatgt ggcacccact agccgttacc atagcaactg cctctttgcc 2280
ccacttaatc ccatcccgtc tgttaaaagg gccctatagt tggaggtggg ggaggtagga 2340
agagcgatga tcacttgtgg actaagtttg ttcgcatccc cttctccaac cccctcagta 2400
catcaccctg ggggaacagg gtccacttgc tcctgggccc acacagtcct gcagtattgt 2460
gtatataagg ccagggcaaa gaggagcagg ttttaaagtg aaaggcaggc aggtgttggg 2520
gaggcagtta ccggggcaac gggaacaggg cgtttcggag gtggttgcca tggggacctg 2580
gatgctgacg aaggctcgcg aggctgtgag cagccacagt gccctgctca gaagccccaa 2640
gctcgtcagt caagccggtt ctccgtttgc actcaggagc acgggcaggc gagtggcccc 2700
tagttctggg ggcagcgaat tccaattggc gcgcctagcc cgggctaggt cgactctgcc 2760
gcggaaaggg gagaagtgtg ggctcctccg agtcgggggc ggactgggac agcacagtcg 2820
gctgagcgca gcgcccccgc cctgcccgcc acgcggcgaa gacgcctgag cgttcgcgcc 2880
cctcgggcga ggaccccacg caagcccgag ccggtcccga ccctggcccc gacgctcgcc 2940
gcccgcccca gccctgaggg cccctctaca atgggcacta gacatgggat ttaatgtcta 3000
tacaatccca tagctaatgc ctgttttaga gaggcgcctc cgccgctcct ttctcatgga 3060
aatggcccgc gagcccgtcc ggcccagcgc ccctcccgcg ggaggaaggc gagcccggcc 3120
cccggcggcc attcgcgccg cggacaaatc cggcgaacaa tgcgcccgcc cagagtgcgg 3180
cccagctgcc gggccgggga tctggccgcg ggacacaaag gggcccgcac gcctctggcg 3240
taccggtctc gagggatccg atctttttcc ctctgccaaa aattatgggg acatcatgaa 3300
gccccttgag catctgactt ctggctaata aaggaaattt attttcattg caatagtgtg 3360
ttggaatttt ttgtgtctct cactcggcgg ccgcccgagt ttaattggtt tatagaactc 3420
ttcaagctag cgaagcaatt cgttgatctg aatttcgacc acccataata cccattaccc 3480
tggtagataa gtagcatggc gggttaatca ttaactacaa ggaaccccta gtgatggagt 3540
tggccactcc ctctctgcgc gctcgctcgc tcactgaggc cgggcgacca aaggtcgccc 3600
gacgcccggg ctttgcccgg gcggcctcag tgagcgagcg agcgcgcag 3649
<210> 788
<211> 3649
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 788
ctgcgcgctc gctcgctcac tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtggccaa ctccatcact 120
aggggttcct tgtagttaat gattaacccg ccatgctact tatctaccag ggtaatgggg 180
atcctctaga tccggtcggg cccgcggtac cgtcgagaag cttgatgtgg gcggagcttc 240
gaaggggcgg gcgcccgtgg ggcgggtcct gagtgggggc gggaccgggg ccggcacctg 300
ggtgaggttc tgcagagggc cctgcgtatg agtgcaagtg ggttttagga ccaggatgag 360
gcggggtggg ggtgcctacc tgacgaccga ccccgaccca ctggacaagc acccaacccc 420
cattccccaa attgcgcatc ccctatcaga gagggggagg ggaaacagga tgcggcgagg 480
cgcgtgcgca ctgccagctt cagcaccgcg gacagtgcct tcgcccccgc ctggcggcgc 540
gcgccaccgc cgcctcagca ctgaaggcgc gctgacgtca ctcgccggtc ccccgcaaac 600
tccccttccc ggccaccttg gtcgcgtccg cgccgccgcc ggcccagccg gaccgcacca 660
cgcgaggcgc gagatagggg ggcacgggcg cgaccatctg cgctgcggcg ccggcgactc 720
agcgctgcct cagtctgcgg tgggcagcgg aggagtcgtg tcgtgcctga gagcgcaggg 780
cgcgcctagc ccgggctagg tcgactcgac tagggataac agggtaattg tttgaatgag 840
gcttcagtac tttacagaat cgttgcctgc acatcttgga aacacttgct gggattactt 900
cttcaggtta acccaacaga aggctaaaga aggtatattg ctgttgacag tgagcgacgt 960
ctcgatatgg agaacccatg ctgtgaagcc acagatgggc atgggtttta tatcgagacg 1020
ctgcctactg cctcggactt caaggggcta ctttaggagc aattatcttg tttactaaaa 1080
ctgaatacct tgctatctct ttgatacatt tttacaaagc tgaattaaaa tggtataaat 1140
tatcacggga tccctgtgcc ttctagttgc cagccatctg ttgtttgccc ctcccccgtg 1200
ccttccttga ccctggaagg tgccactccc actgtccttt cctaataaaa tgaggaaatt 1260
gcatcgcatt gtctgagtag gtgtcattct attctggggg gtggggtggg gcaggacagc 1320
aagggggagg attgggaaga caatagcagg catgctgggg aggcggccgc ccgagtttaa 1380
ttggtttata gaactcttca agctagcact agtgaagcaa ttcgttgcat tatggcctta 1440
ggtcacttca tctccatggg gttcttcttc tgattttcta gaaaatgaga tgggggtgca 1500
gagagcttcc tcagtgacct gcccagggtc acatcagaaa tgtcagagct agaacttgaa 1560
ctcagattac taatcttaaa ttccatgcct tgggggcatg caagtacgat atacagaagg 1620
agtgaactca ttagggcaga tgaccaatga gtttaggaaa gaagagtcca gggcagggta 1680
catctacacc acccgcccag ccctgggtga gtccagccac gttcacctca ttatagttgc 1740
ctctctccag tcctaccttg acgggaagca caagcagaaa ctgggacagg agccccagga 1800
gaccaaatct tcatggtccc tctgggagga tgggtgggga gagctgtggc agaggcctca 1860
ggaggggccc tgctgctcag tggtgacaga taggggtgag aaagcagaca gagtcattcc 1920
gtcagcattc tgggtctgtt tggtacttct tctcacgcta aggtggcggt gtgatatgca 1980
caatggctaa aaagcaggga gagctggaaa gaaacaagga cagagacaga ggccaagtca 2040
accagaccaa ttcccagagg aagcaaagaa accattacag agactacaag ggggaaggga 2100
aggagagatg aattagcttc ccctgtaaac cttagaaccc agctgttgcc agggcaacgg 2160
ggcaatacct gtctcttcag aggagatgaa gttgccaggg taactacatc ctgtctttct 2220
caaggaccat cccagaatgt ggcacccact agccgttacc atagcaactg cctctttgcc 2280
ccacttaatc ccatcccgtc tgttaaaagg gccctatagt tggaggtggg ggaggtagga 2340
agagcgatga tcacttgtgg actaagtttg ttcgcatccc cttctccaac cccctcagta 2400
catcaccctg ggggaacagg gtccacttgc tcctgggccc acacagtcct gcagtattgt 2460
gtatataagg ccagggcaaa gaggagcagg ttttaaagtg aaaggcaggc aggtgttggg 2520
gaggcagtta ccggggcaac gggaacaggg cgtttcggag gtggttgcca tggggacctg 2580
gatgctgacg aaggctcgcg aggctgtgag cagccacagt gccctgctca gaagccccaa 2640
gctcgtcagt caagccggtt ctccgtttgc actcaggagc acgggcaggc gagtggcccc 2700
tagttctggg ggcagcgaat tccaattggc gcgcctagcc cgggctaggt cgactctgcc 2760
gcggaaaggg gagaagtgtg ggctcctccg agtcgggggc ggactgggac agcacagtcg 2820
gctgagcgca gcgcccccgc cctgcccgcc acgcggcgaa gacgcctgag cgttcgcgcc 2880
cctcgggcga ggaccccacg caagcccgag ccggtcccga ccctggcccc gacgctcgcc 2940
gcccgcccca gccctgaggg cccctcgacg tttatctaca acactctgat ttaatgtcta 3000
tacaatcaga gcattgcaga tggactgcga gaggcgcctc cgccgctcct ttctcatgga 3060
aatggcccgc gagcccgtcc ggcccagcgc ccctcccgcg ggaggaaggc gagcccggcc 3120
cccggcggcc attcgcgccg cggacaaatc cggcgaacaa tgcgcccgcc cagagtgcgg 3180
cccagctgcc gggccgggga tctggccgcg ggacacaaag gggcccgcac gcctctggcg 3240
taccggtctc gagggatccg atctttttcc ctctgccaaa aattatgggg acatcatgaa 3300
gccccttgag catctgactt ctggctaata aaggaaattt attttcattg caatagtgtg 3360
ttggaatttt ttgtgtctct cactcggcgg ccgcccgagt ttaattggtt tatagaactc 3420
ttcaagctag cgaagcaatt cgttgatctg aatttcgacc acccataata cccattaccc 3480
tggtagataa gtagcatggc gggttaatca ttaactacaa ggaaccccta gtgatggagt 3540
tggccactcc ctctctgcgc gctcgctcgc tcactgaggc cgggcgacca aaggtcgccc 3600
gacgcccggg ctttgcccgg gcggcctcag tgagcgagcg agcgcgcag 3649
<210> 789
<211> 130
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 789
aggaacccct agtgatggag ttggccactc cctctctgcg cgctcgctcg ctcactgagg 60
ccgggcgacc aaaggtcgcc cgacgcccgg gctttgcccg ggcggcctca gtgagcgagc 120
gagcgcgcag 130
<210> 790
<211> 477
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 790
ctgcagaggg ccctgcgtat gagtgcaagt gggttttagg accaggatga ggcggggtgg 60
gggtgcctac ctgacgaccg accccgaccc actggacaag cacccaaccc ccattcccca 120
aattgcgcat cccctatcag agagggggag gggaaacagg atgcggcgag gcgcgtgcgc 180
actgccagct tcagcaccgc ggacagtgcc ttcgcccccg cctggcggcg cgcgccaccg 240
ccgcctcagc actgaaggcg cgctgacgtc actcgccggt cccccgcaaa ctccccttcc 300
cggccacctt ggtcgcgtcc gcgccgccgc cggcccagcc ggaccgcacc acgcgaggcg 360
cgagataggg gggcacgggc gcgaccatct gcgctgcggc gccggcgact cagcgctgcc 420
tcagtctgcc aattgcagcg gaggagtcgt gtcgtgcctg agagcgcagg gcgcgcc 477
<210> 791
<211> 1357
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 791
cgatcctatc acgagactag cctcgagaag cttgatatca gcacccacat agcagctcac 60
aaatgtctga aactccaatt cttgggaatc tgacacgatc acacatgcag gcaaaatacc 120
aatgtacatg aattaaaaaa aaaaaaaaca acctttaaaa gaaacaaggg ttcagtacca 180
ctactgacat cttgtttccc cagaggcctt actttaatta tttattgttt ccacttagtt 240
gctcaattaa ttaatttaga ggtttttttc ttcctttctt tttctttttt ctttctctct 300
tttttttctt cttaagacag ggtttctctg tgtagctcag gctatcctgg aactcactct 360
gtagaccagg ctggccttgt actcaaagat ctgcctgcct ctgcctcccc agtgctggga 420
ttaaagacat gcaccatcac tgccctgctt tcctcttttt attttgaaaa ttgttcatca 480
acagttacta aacgtgttcg aattccaaga gctgactaga catataagac cattcagcct 540
tctgaataag atgtaggtgt gcccctcctc ttactcctct atttggaagt tggttacttt 600
ctgtatgtag tatgcgaatc cccctctgcc accccgcttt ctgttttaaa acagaaaagg 660
ctgcaacata cagtgtgtgc ttctgttctt gaactggaag cttaggctgt cctggacttg 720
ggttgagacc tgggctcatc cagataggaa atggatttgg tgaccccgcc aggacttcgc 780
aggcaccaca tcgtggtcgt gtgtgggtgc tgtatgcacc cactgattgc gcgcgtgggt 840
tccagagctt ggtggtctgc gagaggagag tgggcaagag tgggtgtgtc tgtggagccc 900
cagctagggg ctgctgcccg ctgctcccac ttgtggctcc tgggcgccgc cagcaggcac 960
atctccggag gacgccgcgg gatgggagct gatgacagga gagcgccgtc tcccgagtga 1020
tggcagcgca cgctgctgcc tcgccgcctc cgccgctcag tcctgatctt acgttagggt 1080
agctgggtac cccctccgcc cgggaaccag ctagtagagg gagaacagag cagagcgtgc 1140
ggcagagccg atcccgcgtc ccgccgaacc ctgccaagcc ccgccaatcc cagcagagca 1200
ggaaccagcg cagctgagcc aacaccggac gccgcactga gacccagcat tccccagccg 1260
ccactacccg gtccccgccg gggtgccggg ctcgtcctgt gagcccctcg tcatgcgtgt 1320
cgggctcttc gactctccag atcagttcca gagcgct 1357
<210> 792
<211> 132
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 792
gatccgatct ttttccctct gccaaaaatt atggggacat catgaagccc cttgagcatc 60
tgacttctgg ctaataaagg aaatttattt tcattgcaat agtgtgttgg aattttttgt 120
gtctctcact cg 132
<210> 793
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 793
tagcaggcat gctggggag 19
<210> 794
<211> 339
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 794
tcgactaggg ataacagggt aattgtttga atgaggcttc agtactttac agaatcgttg 60
cctgcacatc ttggaaacac ttgctgggat tacttcttca ggttaaccca acagaaggct 120
cgagaaggta tattgctgtt gacagtgagc gctaaaacag gcattagcta tgggtagtga 180
agccacagat gcccatagct aatgcctgtt ttattgccta ctgcctcgga attcaagggg 240
ctactttagg agcaattatc ttgtttacta aaactgaata ccttgctatc tctttgatac 300
atttttacaa agctgaatta aaatggtata aattatcac 339
<210> 795
<211> 311
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 795
gtttgaatga ggcttcagta ctttacagaa tcgttgcctg cacatcttgg aaacacttgc 60
tgggattact tcttcaggtt aacccaacag aaggctcgag aaggtatatt gctgttgaca 120
gtgagcgcta aaacaggcat tagctatggg tagtgaagcc acagatgccc atagctaatg 180
cctgttttat tgcctactgc ctcggaattc aaggggctac tttaggagca attatcttgt 240
ttactaaaac tgaatacctt gctatctctt tgatacattt ttacaaagct gaattaaaat 300
ggtataaatt a 311
<210> 796
<211> 1541
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 796
ctgcgcgctc gctcgctcac tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtggccaa ctccatcact 120
aggggttcct tgtagttaat gattaacccg ccatgctact tatctaccag ggtaatgggg 180
atcctctaga tccggtcggg cccgcggtac cgtcgagaag cttgatgtgg gcggagcttc 240
gaaggggcgg gcgcccgtgg ggcgggtcct gagtgggggc gggaccgggg ccggcacctg 300
ggtgaggttc tgcagagggc cctgcgtatg agtgcaagtg ggttttagga ccaggatgag 360
gcggggtggg ggtgcctacc tgacgaccga ccccgaccca ctggacaagc acccaacccc 420
cattccccaa attgcgcatc ccctatcaga gagggggagg ggaaacagga tgcggcgagg 480
cgcgtgcgca ctgccagctt cagcaccgcg gacagtgcct tcgcccccgc ctggcggcgc 540
gcgccaccgc cgcctcagca ctgaaggcgc gctgacgtca ctcgccggtc ccccgcaaac 600
tccccttccc ggccaccttg gtcgcgtccg cgccgccgcc ggcccagccg gaccgcacca 660
cgcgaggcgc gagatagggg ggcacgggcg cgaccatctg cgctgcggcg ccggcgactc 720
agcgctgcct cagtctgcca attgcagcgg aggagtcgtg tcgtgcctga gagcgcaggg 780
cgcgcctagc ccgggctagg tcgactcgac tagggataac agggtaattg tttgaatgag 840
gcttcagtac tttacagaat cgttgcctgc acatcttgga aacacttgct gggattactt 900
cttcaggtta acccaacaga aggctcgaga aggtatattg ctgttgacag tgagcgacat 960
gggttctcca tatcgagact agtgaagcca cagatggtct cgatatggag aacccatgct 1020
gcctactgcc tcggaattca aggggctact ttaggagcaa ttatcttgtt tactaaaact 1080
gaataccttg ctatctcttt gatacatttt tacaaagctg aattaaaatg gtataaatta 1140
tcacgggatc cgatcttttt ccctctgcca aaaattatgg ggacatcatg aagccccttg 1200
agcatctgac ttctggctaa taaaggaaat ttattttcat tgcaatagtg tgttggaatt 1260
ttttgtgtct ctcactcggc ggccgcccga gtttaattgg tttatagaac tcttcaagct 1320
agcgaagcaa ttcgttgatc tgaatttcga ccacccataa tacccattac cctggtagat 1380
aagtagcatg gcgggttaat cattaactac aaggaacccc tagtgatgga gttggccact 1440
ccctctctgc gcgctcgctc gctcactgag gccgggcgac caaaggtcgc ccgacgcccg 1500
ggctttgccc gggcggcctc agtgagcgag cgagcgcgca g 1541
<210> 797
<211> 339
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 797
tcgactaggg ataacagggt aattgtttga atgaggcttc agtactttac agaatcgttg 60
cctgcacatc ttggaaacac ttgctgggat tacttcttca ggttaaccca acagaaggct 120
cgagaaggta tattgctgtt gacagtgagc gacatgggtt ctccatatcg agactagtga 180
agccacagat ggtctcgata tggagaaccc atgctgccta ctgcctcgga attcaagggg 240
ctactttagg agcaattatc ttgtttacta aaactgaata ccttgctatc tctttgatac 300
atttttacaa agctgaatta aaatggtata aattatcac 339
<210> 798
<211> 341
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 798
tcgactaggg ataacagggt aattgtttga atgaggcttc agtactttac agaatcgttg 60
cctgcacatc ttggaaacac ttgctgggat tacttcttca ggttaaccca acagaaggct 120
aaagaaggta tattgctgtt gacagtgagc gacgtctcga tatggagaac ccatgctgtg 180
aagccacaga tgggcatggg ttttatatcg agacgctgcc tactgcctcg gacttcaagg 240
ggctacttta ggagcaatta tcttgtttac taaaactgaa taccttgcta tctctttgat 300
acatttttac aaagctgaat taaaatggta taaattatca c 341
<210> 799
<211> 292
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 799
acgtttccag aacgtctgta gcttttctcc tccttccctc cattttcctc ttggtcttac 60
ctttggccta gtggttggtg tagtgataat gtagcgagat tttctgcaga gcattgcaga 120
tggactgggt tgcgaggtat gagtaaacag tccatacgca atgctccgtg gaacgtcacg 180
cagctttcta cagcatgaca agctgctgag gcttaaatca ggattttcct gtctctttct 240
acaaaatcaa aatgaaaaaa gagggctttt taggcatctc cgagattatg tg 292
<210> 800
<211> 487
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 800
tctgccgcgg aaaggggaga agtgtgggct cctccgagtc gggggcggac tgggacagca 60
cagtcggctg agcgcagcgc ccccgccctg cccgccacgc ggcgaagacg cctgagcgtt 120
cgcgcccctc gggcgaggac cccacgcaag cccgagccgg tcccgaccct ggccccgacg 180
ctcgccgccc gccccagccc tgagggcccc tcgacgttta tctacaacac tctgatttaa 240
tgtctataca atcagagcat tgcagatgga ctgcgagagg cgcctccgcc gctcctttct 300
catggaaatg gcccgcgagc ccgtccggcc cagcgcccct cccgcgggag gaaggcgagc 360
ccggcccccg gcggccattc gcgccgcgga caaatccggc gaacaatgcg cccgcccaga 420
gtgcggccca gctgccgggc cggggatctg gccgcgggac acaaaggggc ccgcacgcct 480
ctggcgt 487
<210> 801
<211> 487
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 801
tctgccgcgg aaaggggaga agtgtgggct cctccgagtc gggggcggac tgggacagca 60
cagtcggctg agcgcagcgc ccccgccctg cccgccacgc ggcgaagacg cctgagcgtt 120
cgcgcccctc gggcgaggac cccacgcaag cccgagccgg tcccgaccct ggccccgacg 180
ctcgccgccc gccccagccc tgagggcccc tctacaatgg gcactagaca tgggatttaa 240
tgtctataca atcccatagc taatgcctgt tttagagagg cgcctccgcc gctcctttct 300
catggaaatg gcccgcgagc ccgtccggcc cagcgcccct cccgcgggag gaaggcgagc 360
ccggcccccg gcggccattc gcgccgcgga caaatccggc gaacaatgcg cccgcccaga 420
gtgcggccca gctgccgggc cggggatctg gccgcgggac acaaaggggc ccgcacgcct 480
ctggcgt 487
<210> 802
<211> 1289
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 802
cattatggcc ttaggtcact tcatctccat ggggttcttc ttctgatttt ctagaaaatg 60
agatgggggt gcagagagct tcctcagtga cctgcccagg gtcacatcag aaatgtcaga 120
gctagaactt gaactcagat tactaatctt aaattccatg ccttgggggc atgcaagtac 180
gatatacaga aggagtgaac tcattagggc agatgaccaa tgagtttagg aaagaagagt 240
ccagggcagg gtacatctac accacccgcc cagccctggg tgagtccagc cacgttcacc 300
tcattatagt tgcctctctc cagtcctacc ttgacgggaa gcacaagcag aaactgggac 360
aggagcccca ggagaccaaa tcttcatggt ccctctggga ggatgggtgg ggagagctgt 420
ggcagaggcc tcaggagggg ccctgctgct cagtggtgac agataggggt gagaaagcag 480
acagagtcat tccgtcagca ttctgggtct gtttggtact tcttctcacg ctaaggtggc 540
ggtgtgatat gcacaatggc taaaaagcag ggagagctgg aaagaaacaa ggacagagac 600
agaggccaag tcaaccagac caattcccag aggaagcaaa gaaaccatta cagagactac 660
aagggggaag ggaaggagag atgaattagc ttcccctgta aaccttagaa cccagctgtt 720
gccagggcaa cggggcaata cctgtctctt cagaggagat gaagttgcca gggtaactac 780
atcctgtctt tctcaaggac catcccagaa tgtggcaccc actagccgtt accatagcaa 840
ctgcctcttt gccccactta atcccatccc gtctgttaaa agggccctat agttggaggt 900
gggggaggta ggaagagcga tgatcacttg tggactaagt ttgttcgcat ccccttctcc 960
aaccccctca gtacatcacc ctgggggaac agggtccact tgctcctggg cccacacagt 1020
cctgcagtat tgtgtatata aggccagggc aaagaggagc aggttttaaa gtgaaaggca 1080
ggcaggtgtt ggggaggcag ttaccggggc aacgggaaca gggcgtttcg gaggtggttg 1140
ccatggggac ctggatgctg acgaaggctc gcgaggctgt gagcagccac agtgccctgc 1200
tcagaagccc caagctcgtc agtcaagccg gttctccgtt tgcactcagg agcacgggca 1260
ggcgagtggc ccctagttct gggggcagc 1289
<210> 803
<211> 2619
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 803
ctgcagaggg ccctgcgtat gagtgcaagt gggttttagg accaggatga ggcggggtgg 60
gggtgcctac ctgacgaccg accccgaccc actggacaag cacccaaccc ccattcccca 120
aattgcgcat cccctatcag agagggggag gggaaacagg atgcggcgag gcgcgtgcgc 180
actgccagct tcagcaccgc ggacagtgcc ttcgcccccg cctggcggcg cgcgccaccg 240
ccgcctcagc actgaaggcg cgctgacgtc actcgccggt cccccgcaaa ctccccttcc 300
cggccacctt ggtcgcgtcc gcgccgccgc cggcccagcc ggaccgcacc acgcgaggcg 360
cgagataggg gggcacgggc gcgaccatct gcgctgcggc gccggcgact cagcgctgcc 420
tcagtctgcg gtgggcagcg gaggagtcgt gtcgtgcctg agagcgcagg gcgcgcctag 480
cccgggctag gtcgactcga ctagggataa cagggtaatt gtttgaatga ggcttcagta 540
ctttacagaa tcgttgcctg cacatcttgg aaacacttgc tgggattact tcttcaggtt 600
aacccaacag aaggctaaag aaggtatatt gctgttgaca gtgagcgacg tctcgatatg 660
gagaacccat gctgtgaagc cacagatggg catgggtttt atatcgagac gctgcctact 720
gcctcggact tcaaggggct actttaggag caattatctt gtttactaaa actgaatacc 780
ttgctatctc tttgatacat ttttacaaag ctgaattaaa atggtataaa ttatcacggg 840
atccgatctt tttccctctg ccaaaaatta tggggacatc atgaagcccc ttgagcatct 900
gacttctggc taataaagga aatttatttt cattgcaata gtgtgttgga attttttgtg 960
tctctcactc ggcggccgca tagtctatcc aggttgagca tcctgctggt ggttacaaga 1020
aactgtttga aactgtggag gaactgtcct cgccgctcac agctcatgta acaggcagga 1080
tccccctctg gctcaccggc agtctccttc gatgtgggcc aggactcttt gaagttggat 1140
ctgagccatt ttaccacctg tttgatgggc aagccctcct gcacaagttt gactttaaag 1200
aaggacatgt cacataccac agaaggttca tccgcactga tgcttacgta cgggcaatga 1260
ctgagaaaag gatcgtcata acagaatttg gcacctgtgc tttcccagat ccctgcaaga 1320
atatattttc caggtttttt tcttactttc gaggagtaga ggttactgac aattgccctt 1380
gttaatgtct acccagtggg ggaagattac tacgcttgca cagagaccaa ctttattaca 1440
aagattaatc cagagacctt ggagacaatt aagcaggttg atctttgcaa ctaagtctct 1500
gtcaatgggg ccactgctca cccccacatt gaaaatgatg gaaccgttta caatattggt 1560
aattgctttg gaaaaaattt ttcaattgcc tacaacattg taaagatccc accactgcaa 1620
gcagacaagg aagatccaat aagcaagtca gagatcgttg tacaattccc ctgcagtgac 1680
cgattcaagc catcttacgt tcatagtttt ggtctgactc ccaactatat cgtttttgtg 1740
gagacaccag tcaaaattaa cctgttcaag ttcctttctt catggagtct ttggggagcc 1800
aactacatgg attgttttga gtccaatgaa accatggggt ttggcttcat attgctgaca 1860
aaaaaaggaa aaagtacctc aataataaat acagaacttc tcctttcaac ctcttccatc 1920
acatcaacac ctatgaagac aatgggtttc tgattgtgga tctctgctgc tggaaaggat 1980
ttgagtttgt ttataattac ttatatttag ccaatttacg tgagaactgg gaagaggtga 2040
aaaaaaatgc cagaaaggct ccccaacctg aagttaggag atatgtactt cctttgaata 2100
ttgacaaggc tgacacaggc aagaatttag tcagctcccc aatacaactg ccactgcaat 2160
tctgtgcagt gacgagacta tctggctgga gcctgaagtt ctcttttcag ggcctcgtca 2220
agcatttgag tttcctcaaa tcaattacca gaagtattgt gggaaacctt acacatatgc 2280
gtatggactt ggcttgaatc actttgttcc agataggctc tgtaagctga atgtcaaaac 2340
taaagaaact tgggtttggc aagagcctga ttcataccca tcagaaccca tctttgtttc 2400
tcacccagat gccttggaag aagatgatgg tgtagttctg agtgtggtgg tgagcccagg 2460
agcaggacaa aagcctgctt atctcctgat tctgaatgcc aaggacttaa gtgaagttgc 2520
ccgggctgaa gtggagatta acatccctgt cacctttcat ggactgttca aaaaatcttg 2580
accggccgcc cgagtttaat tggtttatag aactcttca 2619
<210> 804
<211> 3153
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 804
ctgcgcgctc gctcgctcac tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtggccaa ctccatcact 120
aggggttcct tgtagttaat gattaacccg ccatgctact tatctaccag ggtaatgggg 180
atcctctaga tccggtcggg cccgcggtac cgtcgagaag cttgatgtgg gcggagcttc 240
gaaggggcgg gcgcccgtgg ggcgggtcct gagtgggggc gggaccgggg ccggcacctg 300
ggtgaggttc tgcagagggc cctgcgtatg agtgcaagtg ggttttagga ccaggatgag 360
gcggggtggg ggtgcctacc tgacgaccga ccccgaccca ctggacaagc acccaacccc 420
cattccccaa attgcgcatc ccctatcaga gagggggagg ggaaacagga tgcggcgagg 480
cgcgtgcgca ctgccagctt cagcaccgcg gacagtgcct tcgcccccgc ctggcggcgc 540
gcgccaccgc cgcctcagca ctgaaggcgc gctgacgtca ctcgccggtc ccccgcaaac 600
tccccttccc ggccaccttg gtcgcgtccg cgccgccgcc ggcccagccg gaccgcacca 660
cgcgaggcgc gagatagggg ggcacgggcg cgaccatctg cgctgcggcg ccggcgactc 720
agcgctgcct cagtctgcgg tgggcagcgg aggagtcgtg tcgtgcctga gagcgcaggg 780
cgcgcctagc ccgggctagg tcgactcgac tagggataac agggtaattg tttgaatgag 840
gcttcagtac tttacagaat cgttgcctgc acatcttgga aacacttgct gggattactt 900
cttcaggtta acccaacaga aggctaaaga aggtatattg ctgttgacag tgagcgacgt 960
ctcgatatgg agaacccatg ctgtgaagcc acagatgggc atgggtttta tatcgagacg 1020
ctgcctactg cctcggactt caaggggcta ctttaggagc aattatcttg tttactaaaa 1080
ctgaatacct tgctatctct ttgatacatt tttacaaagc tgaattaaaa tggtataaat 1140
tatcacggga tccgatcttt ttccctctgc caaaaattat ggggacatca tgaagcccct 1200
tgagcatctg acttctggct aataaaggaa atttattttc attgcaatag tgtgttggaa 1260
ttttttgtgt ctctcactcg gcggccgcat agtctatcca ggttgagcat cctgctggtg 1320
gttacaagaa actgtttgaa actgtggagg aactgtcctc gccgctcaca gctcatgtaa 1380
caggcaggat ccccctctgg ctcaccggca gtctccttcg atgtgggcca ggactctttg 1440
aagttggatc tgagccattt taccacctgt ttgatgggca agccctcctg cacaagtttg 1500
actttaaaga aggacatgtc acataccaca gaaggttcat ccgcactgat gcttacgtac 1560
gggcaatgac tgagaaaagg atcgtcataa cagaatttgg cacctgtgct ttcccagatc 1620
cctgcaagaa tatattttcc aggttttttt cttactttcg aggagtagag gttactgaca 1680
attgcccttg ttaatgtcta cccagtgggg gaagattact acgcttgcac agagaccaac 1740
tttattacaa agattaatcc agagaccttg gagacaatta agcaggttga tctttgcaac 1800
taagtctctg tcaatggggc cactgctcac ccccacattg aaaatgatgg aaccgtttac 1860
aatattggta attgctttgg aaaaaatttt tcaattgcct acaacattgt aaagatccca 1920
ccactgcaag cagacaagga agatccaata agcaagtcag agatcgttgt acaattcccc 1980
tgcagtgacc gattcaagcc atcttacgtt catagttttg gtctgactcc caactatatc 2040
gtttttgtgg agacaccagt caaaattaac ctgttcaagt tcctttcttc atggagtctt 2100
tggggagcca actacatgga ttgttttgag tccaatgaaa ccatggggtt tggcttcata 2160
ttgctgacaa aaaaaggaaa aagtacctca ataataaata cagaacttct cctttcaacc 2220
tcttccatca catcaacacc tatgaagaca atgggtttct gattgtggat ctctgctgct 2280
ggaaaggatt tgagtttgtt tataattact tatatttagc caatttacgt gagaactggg 2340
aagaggtgaa aaaaaatgcc agaaaggctc cccaacctga agttaggaga tatgtacttc 2400
ctttgaatat tgacaaggct gacacaggca agaatttagt cagctcccca atacaactgc 2460
cactgcaatt ctgtgcagtg acgagactat ctggctggag cctgaagttc tcttttcagg 2520
gcctcgtcaa gcatttgagt ttcctcaaat caattaccag aagtattgtg ggaaacctta 2580
cacatatgcg tatggacttg gcttgaatca ctttgttcca gataggctct gtaagctgaa 2640
tgtcaaaact aaagaaactt gggtttggca agagcctgat tcatacccat cagaacccat 2700
ctttgtttct cacccagatg ccttggaaga agatgatggt gtagttctga gtgtggtggt 2760
gagcccagga gcaggacaaa agcctgctta tctcctgatt ctgaatgcca aggacttaag 2820
tgaagttgcc cgggctgaag tggagattaa catccctgtc acctttcatg gactgttcaa 2880
aaaatcttga ccggccgccc gagtttaatt ggtttataga actcttcaag ctagcgaagc 2940
aattcgttga tctgaatttc gaccacccat aatacccatt accctggtag ataagtagca 3000
tggcgggtta atcattaact acaaggaacc cctagtgatg gagttggcca ctccctctct 3060
gcgcgctcgc tcgctcactg aggccgggcg accaaaggtc gcccgacgcc cgggctttgc 3120
ccgggcggcc tcagtgagcg agcgagcgcg cag 3153
<210> 805
<211> 2564
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 805
ctgcagaggg ccctgcgtat gagtgcaagt gggttttagg accaggatga ggcggggtgg 60
gggtgcctac ctgacgaccg accccgaccc actggacaag cacccaaccc ccattcccca 120
aattgcgcat cccctatcag agagggggag gggaaacagg atgcggcgag gcgcgtgcgc 180
actgccagct tcagcaccgc ggacagtgcc ttcgcccccg cctggcggcg cgcgccaccg 240
ccgcctcagc actgaaggcg cgctgacgtc actcgccggt cccccgcaaa ctccccttcc 300
cggccacctt ggtcgcgtcc gcgccgccgc cggcccagcc ggaccgcacc acgcgaggcg 360
cgagataggg gggcacgggc gcgaccatct gcgctgcggc gccggcgact cagcgctgcc 420
tcagtctgcg gtgggcagcg gaggagtcgt gtcgtgcctg agagcgcagg gcgcgcctga 480
attcacgttt ccagaacgtc tgtagctttt ctcctccttc cctccatttt cctcttggtc 540
ttacctttgg cctagtggtt ggtgtagtga taatgtagcg agattttctg cagagcattg 600
cagatggact gggttgcgag gtatgagtaa acagtccata cgcaatgctc cgtggaacgt 660
cacgcagctt tctacagcat gacaagctgc tgaggcttaa atcaggattt tcctgtctct 720
ttctacaaaa tcaaaatgaa aaaagagggc tttttaggca tctccgagat tatgtgctcg 780
aggggatccg atctttttcc ctctgccaaa aattatgggg acatcatgaa gccccttgag 840
catctgactt ctggctaata aaggaaattt attttcattg caatagtgtg ttggaatttt 900
ttgtgtctct cactcggcgg ccgcatagtc tatccaggtt gagcatcctg ctggtggtta 960
caagaaactg tttgaaactg tggaggaact gtcctcgccg ctcacagctc atgtaacagg 1020
caggatcccc ctctggctca ccggcagtct ccttcgatgt gggccaggac tctttgaagt 1080
tggatctgag ccattttacc acctgtttga tgggcaagcc ctcctgcaca agtttgactt 1140
taaagaagga catgtcacat accacagaag gttcatccgc actgatgctt acgtacgggc 1200
aatgactgag aaaaggatcg tcataacaga atttggcacc tgtgctttcc cagatccctg 1260
caagaatata ttttccaggt ttttttctta ctttcgagga gtagaggtta ctgacaattg 1320
cccttgttaa tgtctaccca gtgggggaag attactacgc ttgcacagag accaacttta 1380
ttacaaagat taatccagag accttggaga caattaagca ggttgatctt tgcaactaag 1440
tctctgtcaa tggggccact gctcaccccc acattgaaaa tgatggaacc gtttacaata 1500
ttggtaattg ctttggaaaa aatttttcaa ttgcctacaa cattgtaaag atcccaccac 1560
tgcaagcaga caaggaagat ccaataagca agtcagagat cgttgtacaa ttcccctgca 1620
gtgaccgatt caagccatct tacgttcata gttttggtct gactcccaac tatatcgttt 1680
ttgtggagac accagtcaaa attaacctgt tcaagttcct ttcttcatgg agtctttggg 1740
gagccaacta catggattgt tttgagtcca atgaaaccat ggggtttggc ttcatattgc 1800
tgacaaaaaa aggaaaaagt acctcaataa taaatacaga acttctcctt tcaacctctt 1860
ccatcacatc aacacctatg aagacaatgg gtttctgatt gtggatctct gctgctggaa 1920
aggatttgag tttgtttata attacttata tttagccaat ttacgtgaga actgggaaga 1980
ggtgaaaaaa aatgccagaa aggctcccca acctgaagtt aggagatatg tacttccttt 2040
gaatattgac aaggctgaca caggcaagaa tttagtcagc tccccaatac aactgccact 2100
gcaattctgt gcagtgacga gactatctgg ctggagcctg aagttctctt ttcagggcct 2160
cgtcaagcat ttgagtttcc tcaaatcaat taccagaagt attgtgggaa accttacaca 2220
tatgcgtatg gacttggctt gaatcacttt gttccagata ggctctgtaa gctgaatgtc 2280
aaaactaaag aaacttgggt ttggcaagag cctgattcat acccatcaga acccatcttt 2340
gtttctcacc cagatgcctt ggaagaagat gatggtgtag ttctgagtgt ggtggtgagc 2400
ccaggagcag gacaaaagcc tgcttatctc ctgattctga atgccaagga cttaagtgaa 2460
gttgcccggg ctgaagtgga gattaacatc cctgtcacct ttcatggact gttcaaaaaa 2520
tcttgaccgg ccgcccgagt ttaattggtt tatagaactc ttca 2564
<210> 806
<211> 3098
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 806
ctgcgcgctc gctcgctcac tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtggccaa ctccatcact 120
aggggttcct tgtagttaat gattaacccg ccatgctact tatctaccag ggtaatgggg 180
atcctctaga tccggtcggg cccgcggtac cgtcgagaag cttgatgtgg gcggagcttc 240
gaaggggcgg gcgcccgtgg ggcgggtcct gagtgggggc gggaccgggg ccggcacctg 300
ggtgaggttc tgcagagggc cctgcgtatg agtgcaagtg ggttttagga ccaggatgag 360
gcggggtggg ggtgcctacc tgacgaccga ccccgaccca ctggacaagc acccaacccc 420
cattccccaa attgcgcatc ccctatcaga gagggggagg ggaaacagga tgcggcgagg 480
cgcgtgcgca ctgccagctt cagcaccgcg gacagtgcct tcgcccccgc ctggcggcgc 540
gcgccaccgc cgcctcagca ctgaaggcgc gctgacgtca ctcgccggtc ccccgcaaac 600
tccccttccc ggccaccttg gtcgcgtccg cgccgccgcc ggcccagccg gaccgcacca 660
cgcgaggcgc gagatagggg ggcacgggcg cgaccatctg cgctgcggcg ccggcgactc 720
agcgctgcct cagtctgcgg tgggcagcgg aggagtcgtg tcgtgcctga gagcgcaggg 780
cgcgcctgaa ttcacgtttc cagaacgtct gtagcttttc tcctccttcc ctccattttc 840
ctcttggtct tacctttggc ctagtggttg gtgtagtgat aatgtagcga gattttctgc 900
agagcattgc agatggactg ggttgcgagg tatgagtaaa cagtccatac gcaatgctcc 960
gtggaacgtc acgcagcttt ctacagcatg acaagctgct gaggcttaaa tcaggatttt 1020
cctgtctctt tctacaaaat caaaatgaaa aaagagggct ttttaggcat ctccgagatt 1080
atgtgctcga ggggatccga tctttttccc tctgccaaaa attatgggga catcatgaag 1140
ccccttgagc atctgacttc tggctaataa aggaaattta ttttcattgc aatagtgtgt 1200
tggaattttt tgtgtctctc actcggcggc cgcatagtct atccaggttg agcatcctgc 1260
tggtggttac aagaaactgt ttgaaactgt ggaggaactg tcctcgccgc tcacagctca 1320
tgtaacaggc aggatccccc tctggctcac cggcagtctc cttcgatgtg ggccaggact 1380
ctttgaagtt ggatctgagc cattttacca cctgtttgat gggcaagccc tcctgcacaa 1440
gtttgacttt aaagaaggac atgtcacata ccacagaagg ttcatccgca ctgatgctta 1500
cgtacgggca atgactgaga aaaggatcgt cataacagaa tttggcacct gtgctttccc 1560
agatccctgc aagaatatat tttccaggtt tttttcttac tttcgaggag tagaggttac 1620
tgacaattgc ccttgttaat gtctacccag tgggggaaga ttactacgct tgcacagaga 1680
ccaactttat tacaaagatt aatccagaga ccttggagac aattaagcag gttgatcttt 1740
gcaactaagt ctctgtcaat ggggccactg ctcaccccca cattgaaaat gatggaaccg 1800
tttacaatat tggtaattgc tttggaaaaa atttttcaat tgcctacaac attgtaaaga 1860
tcccaccact gcaagcagac aaggaagatc caataagcaa gtcagagatc gttgtacaat 1920
tcccctgcag tgaccgattc aagccatctt acgttcatag ttttggtctg actcccaact 1980
atatcgtttt tgtggagaca ccagtcaaaa ttaacctgtt caagttcctt tcttcatgga 2040
gtctttgggg agccaactac atggattgtt ttgagtccaa tgaaaccatg gggtttggct 2100
tcatattgct gacaaaaaaa ggaaaaagta cctcaataat aaatacagaa cttctccttt 2160
caacctcttc catcacatca acacctatga agacaatggg tttctgattg tggatctctg 2220
ctgctggaaa ggatttgagt ttgtttataa ttacttatat ttagccaatt tacgtgagaa 2280
ctgggaagag gtgaaaaaaa atgccagaaa ggctccccaa cctgaagtta ggagatatgt 2340
acttcctttg aatattgaca aggctgacac aggcaagaat ttagtcagct ccccaataca 2400
actgccactg caattctgtg cagtgacgag actatctggc tggagcctga agttctcttt 2460
tcagggcctc gtcaagcatt tgagtttcct caaatcaatt accagaagta ttgtgggaaa 2520
ccttacacat atgcgtatgg acttggcttg aatcactttg ttccagatag gctctgtaag 2580
ctgaatgtca aaactaaaga aacttgggtt tggcaagagc ctgattcata cccatcagaa 2640
cccatctttg tttctcaccc agatgccttg gaagaagatg atggtgtagt tctgagtgtg 2700
gtggtgagcc caggagcagg acaaaagcct gcttatctcc tgattctgaa tgccaaggac 2760
ttaagtgaag ttgcccgggc tgaagtggag attaacatcc ctgtcacctt tcatggactg 2820
ttcaaaaaat cttgaccggc cgcccgagtt taattggttt atagaactct tcaagctagc 2880
gaagcaattc gttgatctga atttcgacca cccataatac ccattaccct ggtagataag 2940
tagcatggcg ggttaatcat taactacaag gaacccctag tgatggagtt ggccactccc 3000
tctctgcgcg ctcgctcgct cactgaggcc gggcgaccaa aggtcgcccg acgcccgggc 3060
tttgcccggg cggcctcagt gagcgagcga gcgcgcag 3098
<210> 807
<211> 2759
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 807
ctgcagaggg ccctgcgtat gagtgcaagt gggttttagg accaggatga ggcggggtgg 60
gggtgcctac ctgacgaccg accccgaccc actggacaag cacccaaccc ccattcccca 120
aattgcgcat cccctatcag agagggggag gggaaacagg atgcggcgag gcgcgtgcgc 180
actgccagct tcagcaccgc ggacagtgcc ttcgcccccg cctggcggcg cgcgccaccg 240
ccgcctcagc actgaaggcg cgctgacgtc actcgccggt cccccgcaaa ctccccttcc 300
cggccacctt ggtcgcgtcc gcgccgccgc cggcccagcc ggaccgcacc acgcgaggcg 360
cgagataggg gggcacgggc gcgaccatct gcgctgcggc gccggcgact cagcgctgcc 420
tcagtctgcg gtgggcagcg gaggagtcgt gtcgtgcctg agagcgcagg gcgcgcctga 480
attctctgcc gcggaaaggg gagaagtgtg ggctcctccg agtcgggggc ggactgggac 540
agcacagtcg gctgagcgca gcgcccccgc cctgcccgcc acgcggcgaa gacgcctgag 600
cgttcgcgcc cctcgggcga ggaccccacg caagcccgag ccggtcccga ccctggcccc 660
gacgctcgcc gcccgcccca gccctgaggg cccctcgacg tttatctaca acactctgat 720
ttaatgtcta tacaatcaga gcattgcaga tggactgcga gaggcgcctc cgccgctcct 780
ttctcatgga aatggcccgc gagcccgtcc ggcccagcgc ccctcccgcg ggaggaaggc 840
gagcccggcc cccggcggcc attcgcgccg cggacaaatc cggcgaacaa tgcgcccgcc 900
cagagtgcgg cccagctgcc gggccgggga tctggccgcg ggacacaaag gggcccgcac 960
gcctctggcg tctcgagggg atccgatctt tttccctctg ccaaaaatta tggggacatc 1020
atgaagcccc ttgagcatct gacttctggc taataaagga aatttatttt cattgcaata 1080
gtgtgttgga attttttgtg tctctcactc ggcggccgca tagtctatcc aggttgagca 1140
tcctgctggt ggttacaaga aactgtttga aactgtggag gaactgtcct cgccgctcac 1200
agctcatgta acaggcagga tccccctctg gctcaccggc agtctccttc gatgtgggcc 1260
aggactcttt gaagttggat ctgagccatt ttaccacctg tttgatgggc aagccctcct 1320
gcacaagttt gactttaaag aaggacatgt cacataccac agaaggttca tccgcactga 1380
tgcttacgta cgggcaatga ctgagaaaag gatcgtcata acagaatttg gcacctgtgc 1440
tttcccagat ccctgcaaga atatattttc caggtttttt tcttactttc gaggagtaga 1500
ggttactgac aattgccctt gttaatgtct acccagtggg ggaagattac tacgcttgca 1560
cagagaccaa ctttattaca aagattaatc cagagacctt ggagacaatt aagcaggttg 1620
atctttgcaa ctaagtctct gtcaatgggg ccactgctca cccccacatt gaaaatgatg 1680
gaaccgttta caatattggt aattgctttg gaaaaaattt ttcaattgcc tacaacattg 1740
taaagatccc accactgcaa gcagacaagg aagatccaat aagcaagtca gagatcgttg 1800
tacaattccc ctgcagtgac cgattcaagc catcttacgt tcatagtttt ggtctgactc 1860
ccaactatat cgtttttgtg gagacaccag tcaaaattaa cctgttcaag ttcctttctt 1920
catggagtct ttggggagcc aactacatgg attgttttga gtccaatgaa accatggggt 1980
ttggcttcat attgctgaca aaaaaaggaa aaagtacctc aataataaat acagaacttc 2040
tcctttcaac ctcttccatc acatcaacac ctatgaagac aatgggtttc tgattgtgga 2100
tctctgctgc tggaaaggat ttgagtttgt ttataattac ttatatttag ccaatttacg 2160
tgagaactgg gaagaggtga aaaaaaatgc cagaaaggct ccccaacctg aagttaggag 2220
atatgtactt cctttgaata ttgacaaggc tgacacaggc aagaatttag tcagctcccc 2280
aatacaactg ccactgcaat tctgtgcagt gacgagacta tctggctgga gcctgaagtt 2340
ctcttttcag ggcctcgtca agcatttgag tttcctcaaa tcaattacca gaagtattgt 2400
gggaaacctt acacatatgc gtatggactt ggcttgaatc actttgttcc agataggctc 2460
tgtaagctga atgtcaaaac taaagaaact tgggtttggc aagagcctga ttcataccca 2520
tcagaaccca tctttgtttc tcacccagat gccttggaag aagatgatgg tgtagttctg 2580
agtgtggtgg tgagcccagg agcaggacaa aagcctgctt atctcctgat tctgaatgcc 2640
aaggacttaa gtgaagttgc ccgggctgaa gtggagatta acatccctgt cacctttcat 2700
ggactgttca aaaaatcttg accggccgcc cgagtttaat tggtttatag aactcttca 2759
<210> 808
<211> 3293
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 808
ctgcgcgctc gctcgctcac tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtggccaa ctccatcact 120
aggggttcct tgtagttaat gattaacccg ccatgctact tatctaccag ggtaatgggg 180
atcctctaga tccggtcggg cccgcggtac cgtcgagaag cttgatgtgg gcggagcttc 240
gaaggggcgg gcgcccgtgg ggcgggtcct gagtgggggc gggaccgggg ccggcacctg 300
ggtgaggttc tgcagagggc cctgcgtatg agtgcaagtg ggttttagga ccaggatgag 360
gcggggtggg ggtgcctacc tgacgaccga ccccgaccca ctggacaagc acccaacccc 420
cattccccaa attgcgcatc ccctatcaga gagggggagg ggaaacagga tgcggcgagg 480
cgcgtgcgca ctgccagctt cagcaccgcg gacagtgcct tcgcccccgc ctggcggcgc 540
gcgccaccgc cgcctcagca ctgaaggcgc gctgacgtca ctcgccggtc ccccgcaaac 600
tccccttccc ggccaccttg gtcgcgtccg cgccgccgcc ggcccagccg gaccgcacca 660
cgcgaggcgc gagatagggg ggcacgggcg cgaccatctg cgctgcggcg ccggcgactc 720
agcgctgcct cagtctgcgg tgggcagcgg aggagtcgtg tcgtgcctga gagcgcaggg 780
cgcgcctgaa ttctctgccg cggaaagggg agaagtgtgg gctcctccga gtcgggggcg 840
gactgggaca gcacagtcgg ctgagcgcag cgcccccgcc ctgcccgcca cgcggcgaag 900
acgcctgagc gttcgcgccc ctcgggcgag gaccccacgc aagcccgagc cggtcccgac 960
cctggccccg acgctcgccg cccgccccag ccctgagggc ccctcgacgt ttatctacaa 1020
cactctgatt taatgtctat acaatcagag cattgcagat ggactgcgag aggcgcctcc 1080
gccgctcctt tctcatggaa atggcccgcg agcccgtccg gcccagcgcc cctcccgcgg 1140
gaggaaggcg agcccggccc ccggcggcca ttcgcgccgc ggacaaatcc ggcgaacaat 1200
gcgcccgccc agagtgcggc ccagctgccg ggccggggat ctggccgcgg gacacaaagg 1260
ggcccgcacg cctctggcgt ctcgagggga tccgatcttt ttccctctgc caaaaattat 1320
ggggacatca tgaagcccct tgagcatctg acttctggct aataaaggaa atttattttc 1380
attgcaatag tgtgttggaa ttttttgtgt ctctcactcg gcggccgcat agtctatcca 1440
ggttgagcat cctgctggtg gttacaagaa actgtttgaa actgtggagg aactgtcctc 1500
gccgctcaca gctcatgtaa caggcaggat ccccctctgg ctcaccggca gtctccttcg 1560
atgtgggcca ggactctttg aagttggatc tgagccattt taccacctgt ttgatgggca 1620
agccctcctg cacaagtttg actttaaaga aggacatgtc acataccaca gaaggttcat 1680
ccgcactgat gcttacgtac gggcaatgac tgagaaaagg atcgtcataa cagaatttgg 1740
cacctgtgct ttcccagatc cctgcaagaa tatattttcc aggttttttt cttactttcg 1800
aggagtagag gttactgaca attgcccttg ttaatgtcta cccagtgggg gaagattact 1860
acgcttgcac agagaccaac tttattacaa agattaatcc agagaccttg gagacaatta 1920
agcaggttga tctttgcaac taagtctctg tcaatggggc cactgctcac ccccacattg 1980
aaaatgatgg aaccgtttac aatattggta attgctttgg aaaaaatttt tcaattgcct 2040
acaacattgt aaagatccca ccactgcaag cagacaagga agatccaata agcaagtcag 2100
agatcgttgt acaattcccc tgcagtgacc gattcaagcc atcttacgtt catagttttg 2160
gtctgactcc caactatatc gtttttgtgg agacaccagt caaaattaac ctgttcaagt 2220
tcctttcttc atggagtctt tggggagcca actacatgga ttgttttgag tccaatgaaa 2280
ccatggggtt tggcttcata ttgctgacaa aaaaaggaaa aagtacctca ataataaata 2340
cagaacttct cctttcaacc tcttccatca catcaacacc tatgaagaca atgggtttct 2400
gattgtggat ctctgctgct ggaaaggatt tgagtttgtt tataattact tatatttagc 2460
caatttacgt gagaactggg aagaggtgaa aaaaaatgcc agaaaggctc cccaacctga 2520
agttaggaga tatgtacttc ctttgaatat tgacaaggct gacacaggca agaatttagt 2580
cagctcccca atacaactgc cactgcaatt ctgtgcagtg acgagactat ctggctggag 2640
cctgaagttc tcttttcagg gcctcgtcaa gcatttgagt ttcctcaaat caattaccag 2700
aagtattgtg ggaaacctta cacatatgcg tatggacttg gcttgaatca ctttgttcca 2760
gataggctct gtaagctgaa tgtcaaaact aaagaaactt gggtttggca agagcctgat 2820
tcatacccat cagaacccat ctttgtttct cacccagatg ccttggaaga agatgatggt 2880
gtagttctga gtgtggtggt gagcccagga gcaggacaaa agcctgctta tctcctgatt 2940
ctgaatgcca aggacttaag tgaagttgcc cgggctgaag tggagattaa catccctgtc 3000
acctttcatg gactgttcaa aaaatcttga ccggccgccc gagtttaatt ggtttataga 3060
actcttcaag ctagcgaagc aattcgttga tctgaatttc gaccacccat aatacccatt 3120
accctggtag ataagtagca tggcgggtta atcattaact acaaggaacc cctagtgatg 3180
gagttggcca ctccctctct gcgcgctcgc tcgctcactg aggccgggcg accaaaggtc 3240
gcccgacgcc cgggctttgc ccgggcggcc tcagtgagcg agcgagcgcg cag 3293
<210> 809
<211> 2759
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 809
ctgcagaggg ccctgcgtat gagtgcaagt gggttttagg accaggatga ggcggggtgg 60
gggtgcctac ctgacgaccg accccgaccc actggacaag cacccaaccc ccattcccca 120
aattgcgcat cccctatcag agagggggag gggaaacagg atgcggcgag gcgcgtgcgc 180
actgccagct tcagcaccgc ggacagtgcc ttcgcccccg cctggcggcg cgcgccaccg 240
ccgcctcagc actgaaggcg cgctgacgtc actcgccggt cccccgcaaa ctccccttcc 300
cggccacctt ggtcgcgtcc gcgccgccgc cggcccagcc ggaccgcacc acgcgaggcg 360
cgagataggg gggcacgggc gcgaccatct gcgctgcggc gccggcgact cagcgctgcc 420
tcagtctgcg gtgggcagcg gaggagtcgt gtcgtgcctg agagcgcagg gcgcgcctga 480
attctctgcc gcggaaaggg gagaagtgtg ggctcctccg agtcgggggc ggactgggac 540
agcacagtcg gctgagcgca gcgcccccgc cctgcccgcc acgcggcgaa gacgcctgag 600
cgttcgcgcc cctcgggcga ggaccccacg caagcccgag ccggtcccga ccctggcccc 660
gacgctcgcc gcccgcccca gccctgaggg cccctctaca atgggcacta gacatgggat 720
ttaatgtcta tacaatccca tagctaatgc ctgttttaga gaggcgcctc cgccgctcct 780
ttctcatgga aatggcccgc gagcccgtcc ggcccagcgc ccctcccgcg ggaggaaggc 840
gagcccggcc cccggcggcc attcgcgccg cggacaaatc cggcgaacaa tgcgcccgcc 900
cagagtgcgg cccagctgcc gggccgggga tctggccgcg ggacacaaag gggcccgcac 960
gcctctggcg tctcgagggg atccgatctt tttccctctg ccaaaaatta tggggacatc 1020
atgaagcccc ttgagcatct gacttctggc taataaagga aatttatttt cattgcaata 1080
gtgtgttgga attttttgtg tctctcactc ggcggccgca tagtctatcc aggttgagca 1140
tcctgctggt ggttacaaga aactgtttga aactgtggag gaactgtcct cgccgctcac 1200
agctcatgta acaggcagga tccccctctg gctcaccggc agtctccttc gatgtgggcc 1260
aggactcttt gaagttggat ctgagccatt ttaccacctg tttgatgggc aagccctcct 1320
gcacaagttt gactttaaag aaggacatgt cacataccac agaaggttca tccgcactga 1380
tgcttacgta cgggcaatga ctgagaaaag gatcgtcata acagaatttg gcacctgtgc 1440
tttcccagat ccctgcaaga atatattttc caggtttttt tcttactttc gaggagtaga 1500
ggttactgac aattgccctt gttaatgtct acccagtggg ggaagattac tacgcttgca 1560
cagagaccaa ctttattaca aagattaatc cagagacctt ggagacaatt aagcaggttg 1620
atctttgcaa ctaagtctct gtcaatgggg ccactgctca cccccacatt gaaaatgatg 1680
gaaccgttta caatattggt aattgctttg gaaaaaattt ttcaattgcc tacaacattg 1740
taaagatccc accactgcaa gcagacaagg aagatccaat aagcaagtca gagatcgttg 1800
tacaattccc ctgcagtgac cgattcaagc catcttacgt tcatagtttt ggtctgactc 1860
ccaactatat cgtttttgtg gagacaccag tcaaaattaa cctgttcaag ttcctttctt 1920
catggagtct ttggggagcc aactacatgg attgttttga gtccaatgaa accatggggt 1980
ttggcttcat attgctgaca aaaaaaggaa aaagtacctc aataataaat acagaacttc 2040
tcctttcaac ctcttccatc acatcaacac ctatgaagac aatgggtttc tgattgtgga 2100
tctctgctgc tggaaaggat ttgagtttgt ttataattac ttatatttag ccaatttacg 2160
tgagaactgg gaagaggtga aaaaaaatgc cagaaaggct ccccaacctg aagttaggag 2220
atatgtactt cctttgaata ttgacaaggc tgacacaggc aagaatttag tcagctcccc 2280
aatacaactg ccactgcaat tctgtgcagt gacgagacta tctggctgga gcctgaagtt 2340
ctcttttcag ggcctcgtca agcatttgag tttcctcaaa tcaattacca gaagtattgt 2400
gggaaacctt acacatatgc gtatggactt ggcttgaatc actttgttcc agataggctc 2460
tgtaagctga atgtcaaaac taaagaaact tgggtttggc aagagcctga ttcataccca 2520
tcagaaccca tctttgtttc tcacccagat gccttggaag aagatgatgg tgtagttctg 2580
agtgtggtgg tgagcccagg agcaggacaa aagcctgctt atctcctgat tctgaatgcc 2640
aaggacttaa gtgaagttgc ccgggctgaa gtggagatta acatccctgt cacctttcat 2700
ggactgttca aaaaatcttg accggccgcc cgagtttaat tggtttatag aactcttca 2759
<210> 810
<211> 3293
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 810
ctgcgcgctc gctcgctcac tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtggccaa ctccatcact 120
aggggttcct tgtagttaat gattaacccg ccatgctact tatctaccag ggtaatgggg 180
atcctctaga tccggtcggg cccgcggtac cgtcgagaag cttgatgtgg gcggagcttc 240
gaaggggcgg gcgcccgtgg ggcgggtcct gagtgggggc gggaccgggg ccggcacctg 300
ggtgaggttc tgcagagggc cctgcgtatg agtgcaagtg ggttttagga ccaggatgag 360
gcggggtggg ggtgcctacc tgacgaccga ccccgaccca ctggacaagc acccaacccc 420
cattccccaa attgcgcatc ccctatcaga gagggggagg ggaaacagga tgcggcgagg 480
cgcgtgcgca ctgccagctt cagcaccgcg gacagtgcct tcgcccccgc ctggcggcgc 540
gcgccaccgc cgcctcagca ctgaaggcgc gctgacgtca ctcgccggtc ccccgcaaac 600
tccccttccc ggccaccttg gtcgcgtccg cgccgccgcc ggcccagccg gaccgcacca 660
cgcgaggcgc gagatagggg ggcacgggcg cgaccatctg cgctgcggcg ccggcgactc 720
agcgctgcct cagtctgcgg tgggcagcgg aggagtcgtg tcgtgcctga gagcgcaggg 780
cgcgcctgaa ttctctgccg cggaaagggg agaagtgtgg gctcctccga gtcgggggcg 840
gactgggaca gcacagtcgg ctgagcgcag cgcccccgcc ctgcccgcca cgcggcgaag 900
acgcctgagc gttcgcgccc ctcgggcgag gaccccacgc aagcccgagc cggtcccgac 960
cctggccccg acgctcgccg cccgccccag ccctgagggc ccctctacaa tgggcactag 1020
acatgggatt taatgtctat acaatcccat agctaatgcc tgttttagag aggcgcctcc 1080
gccgctcctt tctcatggaa atggcccgcg agcccgtccg gcccagcgcc cctcccgcgg 1140
gaggaaggcg agcccggccc ccggcggcca ttcgcgccgc ggacaaatcc ggcgaacaat 1200
gcgcccgccc agagtgcggc ccagctgccg ggccggggat ctggccgcgg gacacaaagg 1260
ggcccgcacg cctctggcgt ctcgagggga tccgatcttt ttccctctgc caaaaattat 1320
ggggacatca tgaagcccct tgagcatctg acttctggct aataaaggaa atttattttc 1380
attgcaatag tgtgttggaa ttttttgtgt ctctcactcg gcggccgcat agtctatcca 1440
ggttgagcat cctgctggtg gttacaagaa actgtttgaa actgtggagg aactgtcctc 1500
gccgctcaca gctcatgtaa caggcaggat ccccctctgg ctcaccggca gtctccttcg 1560
atgtgggcca ggactctttg aagttggatc tgagccattt taccacctgt ttgatgggca 1620
agccctcctg cacaagtttg actttaaaga aggacatgtc acataccaca gaaggttcat 1680
ccgcactgat gcttacgtac gggcaatgac tgagaaaagg atcgtcataa cagaatttgg 1740
cacctgtgct ttcccagatc cctgcaagaa tatattttcc aggttttttt cttactttcg 1800
aggagtagag gttactgaca attgcccttg ttaatgtcta cccagtgggg gaagattact 1860
acgcttgcac agagaccaac tttattacaa agattaatcc agagaccttg gagacaatta 1920
agcaggttga tctttgcaac taagtctctg tcaatggggc cactgctcac ccccacattg 1980
aaaatgatgg aaccgtttac aatattggta attgctttgg aaaaaatttt tcaattgcct 2040
acaacattgt aaagatccca ccactgcaag cagacaagga agatccaata agcaagtcag 2100
agatcgttgt acaattcccc tgcagtgacc gattcaagcc atcttacgtt catagttttg 2160
gtctgactcc caactatatc gtttttgtgg agacaccagt caaaattaac ctgttcaagt 2220
tcctttcttc atggagtctt tggggagcca actacatgga ttgttttgag tccaatgaaa 2280
ccatggggtt tggcttcata ttgctgacaa aaaaaggaaa aagtacctca ataataaata 2340
cagaacttct cctttcaacc tcttccatca catcaacacc tatgaagaca atgggtttct 2400
gattgtggat ctctgctgct ggaaaggatt tgagtttgtt tataattact tatatttagc 2460
caatttacgt gagaactggg aagaggtgaa aaaaaatgcc agaaaggctc cccaacctga 2520
agttaggaga tatgtacttc ctttgaatat tgacaaggct gacacaggca agaatttagt 2580
cagctcccca atacaactgc cactgcaatt ctgtgcagtg acgagactat ctggctggag 2640
cctgaagttc tcttttcagg gcctcgtcaa gcatttgagt ttcctcaaat caattaccag 2700
aagtattgtg ggaaacctta cacatatgcg tatggacttg gcttgaatca ctttgttcca 2760
gataggctct gtaagctgaa tgtcaaaact aaagaaactt gggtttggca agagcctgat 2820
tcatacccat cagaacccat ctttgtttct cacccagatg ccttggaaga agatgatggt 2880
gtagttctga gtgtggtggt gagcccagga gcaggacaaa agcctgctta tctcctgatt 2940
ctgaatgcca aggacttaag tgaagttgcc cgggctgaag tggagattaa catccctgtc 3000
acctttcatg gactgttcaa aaaatcttga ccggccgccc gagtttaatt ggtttataga 3060
actcttcaag ctagcgaagc aattcgttga tctgaatttc gaccacccat aatacccatt 3120
accctggtag ataagtagca tggcgggtta atcattaact acaaggaacc cctagtgatg 3180
gagttggcca ctccctctct gcgcgctcgc tcgctcactg aggccgggcg accaaaggtc 3240
gcccgacgcc cgggctttgc ccgggcggcc tcagtgagcg agcgagcgcg cag 3293
<210> 811
<211> 2892
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 811
ctgcagaggg ccctgcgtat gagtgcaagt gggttttagg accaggatga ggcggggtgg 60
gggtgcctac ctgacgaccg accccgaccc actggacaag cacccaaccc ccattcccca 120
aattgcgcat cccctatcag agagggggag gggaaacagg atgcggcgag gcgcgtgcgc 180
actgccagct tcagcaccgc ggacagtgcc ttcgcccccg cctggcggcg cgcgccaccg 240
ccgcctcagc actgaaggcg cgctgacgtc actcgccggt cccccgcaaa ctccccttcc 300
cggccacctt ggtcgcgtcc gcgccgccgc cggcccagcc ggaccgcacc acgcgaggcg 360
cgagataggg gggcacgggc gcgaccatct gcgctgcggc gccggcgact cagcgctgcc 420
tcagtctgcg gtgggcagcg gaggagtcgt gtcgtgcctg agagcgcagg gcgcgcctag 480
cccgggctag gtcgactcga ctagggataa cagggtaatt gtttgaatga ggcttcagta 540
ctttacagaa tcgttgcctg cacatcttgg aaacacttgc tgggattact tcttcaggtt 600
aacccaacag aaggctaaag aaggtatatt gctgttgaca gtgagcgacg tctcgatatg 660
gagaacccat gctgtgaagc cacagatggg catgggtttt atatcgagac gctgcctact 720
gcctcggact tcaaggggct actttaggag caattatctt gtttactaaa actgaatacc 780
ttgctatctc tttgatacat ttttacaaag ctgaattaaa atggtataaa ttatcacggg 840
atccgaattc acgtttccag aacgtctgta gcttttctcc tccttccctc cattttcctc 900
ttggtcttac ctttggccta gtggttggtg tagtgataat gtagcgagat tttctgcaga 960
gcattgcaga tggactgggt tgcgaggtat gagtaaacag tccatacgca atgctccgtg 1020
gaacgtcacg cagctttcta cagcatgaca agctgctgag gcttaaatca ggattttcct 1080
gtctctttct acaaaatcaa aatgaaaaaa gagggctttt taggcatctc cgagattatg 1140
tgctcgaggg gatccgatct ttttccctct gccaaaaatt atggggacat catgaagccc 1200
cttgagcatc tgacttctgg ctaataaagg aaatttattt tcattgcaat agtgtgttgg 1260
aattttttgt gtctctcact cggcggccgc atagtctatc caggttgagc atcctgctgg 1320
tggttacaag aaactgtttg aaactgtgga ggaactgtcc tcgccgctca cagctcatgt 1380
aacaggcagg atccccctct ggctcaccgg cagtctcctt cgatgtgggc caggactctt 1440
tgaagttgga tctgagccat tttaccacct gtttgatggg caagccctcc tgcacaagtt 1500
tgactttaaa gaaggacatg tcacatacca cagaaggttc atccgcactg atgcttacgt 1560
acgggcaatg actgagaaaa ggatcgtcat aacagaattt ggcacctgtg ctttcccaga 1620
tccctgcaag aatatatttt ccaggttttt ttcttacttt cgaggagtag aggttactga 1680
caattgccct tgttaatgtc tacccagtgg gggaagatta ctacgcttgc acagagacca 1740
actttattac aaagattaat ccagagacct tggagacaat taagcaggtt gatctttgca 1800
actaagtctc tgtcaatggg gccactgctc acccccacat tgaaaatgat ggaaccgttt 1860
acaatattgg taattgcttt ggaaaaaatt tttcaattgc ctacaacatt gtaaagatcc 1920
caccactgca agcagacaag gaagatccaa taagcaagtc agagatcgtt gtacaattcc 1980
cctgcagtga ccgattcaag ccatcttacg ttcatagttt tggtctgact cccaactata 2040
tcgtttttgt ggagacacca gtcaaaatta acctgttcaa gttcctttct tcatggagtc 2100
tttggggagc caactacatg gattgttttg agtccaatga aaccatgggg tttggcttca 2160
tattgctgac aaaaaaagga aaaagtacct caataataaa tacagaactt ctcctttcaa 2220
cctcttccat cacatcaaca cctatgaaga caatgggttt ctgattgtgg atctctgctg 2280
ctggaaagga tttgagtttg tttataatta cttatattta gccaatttac gtgagaactg 2340
ggaagaggtg aaaaaaaatg ccagaaaggc tccccaacct gaagttagga gatatgtact 2400
tcctttgaat attgacaagg ctgacacagg caagaattta gtcagctccc caatacaact 2460
gccactgcaa ttctgtgcag tgacgagact atctggctgg agcctgaagt tctcttttca 2520
gggcctcgtc aagcatttga gtttcctcaa atcaattacc agaagtattg tgggaaacct 2580
tacacatatg cgtatggact tggcttgaat cactttgttc cagataggct ctgtaagctg 2640
aatgtcaaaa ctaaagaaac ttgggtttgg caagagcctg attcataccc atcagaaccc 2700
atctttgttt ctcacccaga tgccttggaa gaagatgatg gtgtagttct gagtgtggtg 2760
gtgagcccag gagcaggaca aaagcctgct tatctcctga ttctgaatgc caaggactta 2820
agtgaagttg cccgggctga agtggagatt aacatccctg tcacctttca tggactgttc 2880
aaaaaatctt ga 2892
<210> 812
<211> 3432
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 812
ctgcgcgctc gctcgctcac tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtggccaa ctccatcact 120
aggggttcct tgtagttaat gattaacccg ccatgctact tatctaccag ggtaatgggg 180
atcctctaga tccggtcggg cccgcggtac cgtcgagaag cttgatgtgg gcggagcttc 240
gaaggggcgg gcgcccgtgg ggcgggtcct gagtgggggc gggaccgggg ccggcacctg 300
ggtgaggttc tgcagagggc cctgcgtatg agtgcaagtg ggttttagga ccaggatgag 360
gcggggtggg ggtgcctacc tgacgaccga ccccgaccca ctggacaagc acccaacccc 420
cattccccaa attgcgcatc ccctatcaga gagggggagg ggaaacagga tgcggcgagg 480
cgcgtgcgca ctgccagctt cagcaccgcg gacagtgcct tcgcccccgc ctggcggcgc 540
gcgccaccgc cgcctcagca ctgaaggcgc gctgacgtca ctcgccggtc ccccgcaaac 600
tccccttccc ggccaccttg gtcgcgtccg cgccgccgcc ggcccagccg gaccgcacca 660
cgcgaggcgc gagatagggg ggcacgggcg cgaccatctg cgctgcggcg ccggcgactc 720
agcgctgcct cagtctgcgg tgggcagcgg aggagtcgtg tcgtgcctga gagcgcaggg 780
cgcgcctagc ccgggctagg tcgactcgac tagggataac agggtaattg tttgaatgag 840
gcttcagtac tttacagaat cgttgcctgc acatcttgga aacacttgct gggattactt 900
cttcaggtta acccaacaga aggctaaaga aggtatattg ctgttgacag tgagcgacgt 960
ctcgatatgg agaacccatg ctgtgaagcc acagatgggc atgggtttta tatcgagacg 1020
ctgcctactg cctcggactt caaggggcta ctttaggagc aattatcttg tttactaaaa 1080
ctgaatacct tgctatctct ttgatacatt tttacaaagc tgaattaaaa tggtataaat 1140
tatcacggga tccgaattca cgtttccaga acgtctgtag cttttctcct ccttccctcc 1200
attttcctct tggtcttacc tttggcctag tggttggtgt agtgataatg tagcgagatt 1260
ttctgcagag cattgcagat ggactgggtt gcgaggtatg agtaaacagt ccatacgcaa 1320
tgctccgtgg aacgtcacgc agctttctac agcatgacaa gctgctgagg cttaaatcag 1380
gattttcctg tctctttcta caaaatcaaa atgaaaaaag agggcttttt aggcatctcc 1440
gagattatgt gctcgagggg atccgatctt tttccctctg ccaaaaatta tggggacatc 1500
atgaagcccc ttgagcatct gacttctggc taataaagga aatttatttt cattgcaata 1560
gtgtgttgga attttttgtg tctctcactc ggcggccgca tagtctatcc aggttgagca 1620
tcctgctggt ggttacaaga aactgtttga aactgtggag gaactgtcct cgccgctcac 1680
agctcatgta acaggcagga tccccctctg gctcaccggc agtctccttc gatgtgggcc 1740
aggactcttt gaagttggat ctgagccatt ttaccacctg tttgatgggc aagccctcct 1800
gcacaagttt gactttaaag aaggacatgt cacataccac agaaggttca tccgcactga 1860
tgcttacgta cgggcaatga ctgagaaaag gatcgtcata acagaatttg gcacctgtgc 1920
tttcccagat ccctgcaaga atatattttc caggtttttt tcttactttc gaggagtaga 1980
ggttactgac aattgccctt gttaatgtct acccagtggg ggaagattac tacgcttgca 2040
cagagaccaa ctttattaca aagattaatc cagagacctt ggagacaatt aagcaggttg 2100
atctttgcaa ctaagtctct gtcaatgggg ccactgctca cccccacatt gaaaatgatg 2160
gaaccgttta caatattggt aattgctttg gaaaaaattt ttcaattgcc tacaacattg 2220
taaagatccc accactgcaa gcagacaagg aagatccaat aagcaagtca gagatcgttg 2280
tacaattccc ctgcagtgac cgattcaagc catcttacgt tcatagtttt ggtctgactc 2340
ccaactatat cgtttttgtg gagacaccag tcaaaattaa cctgttcaag ttcctttctt 2400
catggagtct ttggggagcc aactacatgg attgttttga gtccaatgaa accatggggt 2460
ttggcttcat attgctgaca aaaaaaggaa aaagtacctc aataataaat acagaacttc 2520
tcctttcaac ctcttccatc acatcaacac ctatgaagac aatgggtttc tgattgtgga 2580
tctctgctgc tggaaaggat ttgagtttgt ttataattac ttatatttag ccaatttacg 2640
tgagaactgg gaagaggtga aaaaaaatgc cagaaaggct ccccaacctg aagttaggag 2700
atatgtactt cctttgaata ttgacaaggc tgacacaggc aagaatttag tcagctcccc 2760
aatacaactg ccactgcaat tctgtgcagt gacgagacta tctggctgga gcctgaagtt 2820
ctcttttcag ggcctcgtca agcatttgag tttcctcaaa tcaattacca gaagtattgt 2880
gggaaacctt acacatatgc gtatggactt ggcttgaatc actttgttcc agataggctc 2940
tgtaagctga atgtcaaaac taaagaaact tgggtttggc aagagcctga ttcataccca 3000
tcagaaccca tctttgtttc tcacccagat gccttggaag aagatgatgg tgtagttctg 3060
agtgtggtgg tgagcccagg agcaggacaa aagcctgctt atctcctgat tctgaatgcc 3120
aaggacttaa gtgaagttgc ccgggctgaa gtggagatta acatccctgt cacctttcat 3180
ggactgttca aaaaatcttg accggccggc tagcgaagca attcgttgat ctgaatttcg 3240
accacccata atacccatta ccctggtaga taagtagcat ggcgggttaa tcattaacta 3300
caaggaaccc ctagtgatgg agttggccac tccctctctg cgcgctcgct cgctcactga 3360
ggccgggcga ccaaaggtcg cccgacgccc gggctttgcc cgggcggcct cagtgagcga 3420
gcgagcgcgc ag 3432
<210> 813
<211> 2920
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 813
ctgcagaggg ccctgcgtat gagtgcaagt gggttttagg accaggatga ggcggggtgg 60
gggtgcctac ctgacgaccg accccgaccc actggacaag cacccaaccc ccattcccca 120
aattgcgcat cccctatcag agagggggag gggaaacagg atgcggcgag gcgcgtgcgc 180
actgccagct tcagcaccgc ggacagtgcc ttcgcccccg cctggcggcg cgcgccaccg 240
ccgcctcagc actgaaggcg cgctgacgtc actcgccggt cccccgcaaa ctccccttcc 300
cggccacctt ggtcgcgtcc gcgccgccgc cggcccagcc ggaccgcacc acgcgaggcg 360
cgagataggg gggcacgggc gcgaccatct gcgctgcggc gccggcgact cagcgctgcc 420
tcagtctgcg gtgggcagcg gaggagtcgt gtcgtgcctg agagcgcagg gcgcgcctag 480
cccgggctag gtcgactcga ctagggataa cagggtaatt gtttgaatga ggcttcagta 540
ctttacagaa tcgttgcctg cacatcttgg aaacacttgc tgggattact tcttcaggtt 600
aacccaacag aaggctaaag aaggtatatt gctgttgaca gtgagcgacg tctcgatatg 660
gagaacccat gctgtgaagc cacagatggg catgggtttt atatcgagac gctgcctact 720
gcctcggact tcaaggggct actttaggag caattatctt gtttactaaa actgaatacc 780
ttgctatctc tttgatacat ttttacaaag ctgaattaaa atggtataaa ttatcacggg 840
atccctgtgc cttctagttg ccagccatct gttgtttgcc cctcccccgt gccttccttg 900
accctggaag gtgccactcc cactgtcctt tcctaataaa atgaggaaat tgcatcgcat 960
tgtctgagta ggtgtcattc tattctgggg ggtggggtgg ggcaggacag caagggggag 1020
gattgggaag acaatagcag gcatgctggg gaggcggccg cccgagttta attggtttat 1080
agaactcttc aagctagcac tagtgaagca attcgttgca ttatggcctt aggtcacttc 1140
atctccatgg ggttcttctt ctgattttct agaaaatgag atgggggtgc agagagcttc 1200
ctcagtgacc tgcccagggt cacatcagaa atgtcagagc tagaacttga actcagatta 1260
ctaatcttaa attccatgcc ttgggggcat gcaagtacga tatacagaag gagtgaactc 1320
attagggcag atgaccaatg agtttaggaa agaagagtcc agggcagggt acatctacac 1380
cacccgccca gccctgggtg agtccagcca cgttcacctc attatagttg cctctctcca 1440
gtcctacctt gacgggaagc acaagcagaa actgggacag gagccccagg agaccaaatc 1500
ttcatggtcc ctctgggagg atgggtgggg agagctgtgg cagaggcctc aggaggggcc 1560
ctgctgctca gtggtgacag ataggggtga gaaagcagac agagtcattc cgtcagcatt 1620
ctgggtctgt ttggtacttc ttctcacgct aaggtggcgg tgtgatatgc acaatggcta 1680
aaaagcaggg agagctggaa agaaacaagg acagagacag aggccaagtc aaccagacca 1740
attcccagag gaagcaaaga aaccattaca gagactacaa gggggaaggg aaggagagat 1800
gaattagctt cccctgtaaa ccttagaacc cagctgttgc cagggcaacg gggcaatacc 1860
tgtctcttca gaggagatga agttgccagg gtaactacat cctgtctttc tcaaggacca 1920
tcccagaatg tggcacccac tagccgttac catagcaact gcctctttgc cccacttaat 1980
cccatcccgt ctgttaaaag ggccctatag ttggaggtgg gggaggtagg aagagcgatg 2040
atcacttgtg gactaagttt gttcgcatcc ccttctccaa ccccctcagt acatcaccct 2100
gggggaacag ggtccacttg ctcctgggcc cacacagtcc tgcagtattg tgtatataag 2160
gccagggcaa agaggagcag gttttaaagt gaaaggcagg caggtgttgg ggaggcagtt 2220
accggggcaa cgggaacagg gcgtttcgga ggtggttgcc atggggacct ggatgctgac 2280
gaaggctcgc gaggctgtga gcagccacag tgccctgctc agaagcccca agctcgtcag 2340
tcaagccggt tctccgtttg cactcaggag cacgggcagg cgagtggccc ctagttctgg 2400
gggcagcgaa ttccaattgg cgcgcctagc ccgggctagg tcgacacgtt tccagaacgt 2460
ctgtagcttt tctcctcctt ccctccattt tcctcttggt cttacctttg gcctagtggt 2520
tggtgtagtg ataatgtagc gagattttct gcagagcatt gcagatggac tgggttgcga 2580
ggtatgagta aacagtccat acgcaatgct ccgtggaacg tcacgcagct ttctacagca 2640
tgacaagctg ctgaggctta aatcaggatt ttcctgtctc tttctacaaa atcaaaatga 2700
aaaaagaggg ctttttaggc atctccgaga ttatgtgacc ggtctcgagg gatccgatct 2760
ttttccctct gccaaaaatt atggggacat catgaagccc cttgagcatc tgacttctgg 2820
ctaataaagg aaatttattt tcattgcaat agtgtgttgg aattttttgt gtctctcact 2880
cggcggccgc ccgagtttaa ttggtttata gaactcttca 2920
<210> 814
<211> 3454
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 814
ctgcgcgctc gctcgctcac tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtggccaa ctccatcact 120
aggggttcct tgtagttaat gattaacccg ccatgctact tatctaccag ggtaatgggg 180
atcctctaga tccggtcggg cccgcggtac cgtcgagaag cttgatgtgg gcggagcttc 240
gaaggggcgg gcgcccgtgg ggcgggtcct gagtgggggc gggaccgggg ccggcacctg 300
ggtgaggttc tgcagagggc cctgcgtatg agtgcaagtg ggttttagga ccaggatgag 360
gcggggtggg ggtgcctacc tgacgaccga ccccgaccca ctggacaagc acccaacccc 420
cattccccaa attgcgcatc ccctatcaga gagggggagg ggaaacagga tgcggcgagg 480
cgcgtgcgca ctgccagctt cagcaccgcg gacagtgcct tcgcccccgc ctggcggcgc 540
gcgccaccgc cgcctcagca ctgaaggcgc gctgacgtca ctcgccggtc ccccgcaaac 600
tccccttccc ggccaccttg gtcgcgtccg cgccgccgcc ggcccagccg gaccgcacca 660
cgcgaggcgc gagatagggg ggcacgggcg cgaccatctg cgctgcggcg ccggcgactc 720
agcgctgcct cagtctgcgg tgggcagcgg aggagtcgtg tcgtgcctga gagcgcaggg 780
cgcgcctagc ccgggctagg tcgactcgac tagggataac agggtaattg tttgaatgag 840
gcttcagtac tttacagaat cgttgcctgc acatcttgga aacacttgct gggattactt 900
cttcaggtta acccaacaga aggctaaaga aggtatattg ctgttgacag tgagcgacgt 960
ctcgatatgg agaacccatg ctgtgaagcc acagatgggc atgggtttta tatcgagacg 1020
ctgcctactg cctcggactt caaggggcta ctttaggagc aattatcttg tttactaaaa 1080
ctgaatacct tgctatctct ttgatacatt tttacaaagc tgaattaaaa tggtataaat 1140
tatcacggga tccctgtgcc ttctagttgc cagccatctg ttgtttgccc ctcccccgtg 1200
ccttccttga ccctggaagg tgccactccc actgtccttt cctaataaaa tgaggaaatt 1260
gcatcgcatt gtctgagtag gtgtcattct attctggggg gtggggtggg gcaggacagc 1320
aagggggagg attgggaaga caatagcagg catgctgggg aggcggccgc ccgagtttaa 1380
ttggtttata gaactcttca agctagcact agtgaagcaa ttcgttgcat tatggcctta 1440
ggtcacttca tctccatggg gttcttcttc tgattttcta gaaaatgaga tgggggtgca 1500
gagagcttcc tcagtgacct gcccagggtc acatcagaaa tgtcagagct agaacttgaa 1560
ctcagattac taatcttaaa ttccatgcct tgggggcatg caagtacgat atacagaagg 1620
agtgaactca ttagggcaga tgaccaatga gtttaggaaa gaagagtcca gggcagggta 1680
catctacacc acccgcccag ccctgggtga gtccagccac gttcacctca ttatagttgc 1740
ctctctccag tcctaccttg acgggaagca caagcagaaa ctgggacagg agccccagga 1800
gaccaaatct tcatggtccc tctgggagga tgggtgggga gagctgtggc agaggcctca 1860
ggaggggccc tgctgctcag tggtgacaga taggggtgag aaagcagaca gagtcattcc 1920
gtcagcattc tgggtctgtt tggtacttct tctcacgcta aggtggcggt gtgatatgca 1980
caatggctaa aaagcaggga gagctggaaa gaaacaagga cagagacaga ggccaagtca 2040
accagaccaa ttcccagagg aagcaaagaa accattacag agactacaag ggggaaggga 2100
aggagagatg aattagcttc ccctgtaaac cttagaaccc agctgttgcc agggcaacgg 2160
ggcaatacct gtctcttcag aggagatgaa gttgccaggg taactacatc ctgtctttct 2220
caaggaccat cccagaatgt ggcacccact agccgttacc atagcaactg cctctttgcc 2280
ccacttaatc ccatcccgtc tgttaaaagg gccctatagt tggaggtggg ggaggtagga 2340
agagcgatga tcacttgtgg actaagtttg ttcgcatccc cttctccaac cccctcagta 2400
catcaccctg ggggaacagg gtccacttgc tcctgggccc acacagtcct gcagtattgt 2460
gtatataagg ccagggcaaa gaggagcagg ttttaaagtg aaaggcaggc aggtgttggg 2520
gaggcagtta ccggggcaac gggaacaggg cgtttcggag gtggttgcca tggggacctg 2580
gatgctgacg aaggctcgcg aggctgtgag cagccacagt gccctgctca gaagccccaa 2640
gctcgtcagt caagccggtt ctccgtttgc actcaggagc acgggcaggc gagtggcccc 2700
tagttctggg ggcagcgaat tccaattggc gcgcctagcc cgggctaggt cgacacgttt 2760
ccagaacgtc tgtagctttt ctcctccttc cctccatttt cctcttggtc ttacctttgg 2820
cctagtggtt ggtgtagtga taatgtagcg agattttctg cagagcattg cagatggact 2880
gggttgcgag gtatgagtaa acagtccata cgcaatgctc cgtggaacgt cacgcagctt 2940
tctacagcat gacaagctgc tgaggcttaa atcaggattt tcctgtctct ttctacaaaa 3000
tcaaaatgaa aaaagagggc tttttaggca tctccgagat tatgtgaccg gtctcgaggg 3060
atccgatctt tttccctctg ccaaaaatta tggggacatc atgaagcccc ttgagcatct 3120
gacttctggc taataaagga aatttatttt cattgcaata gtgtgttgga attttttgtg 3180
tctctcactc ggcggccgcc cgagtttaat tggtttatag aactcttcaa gctagcgaag 3240
caattcgttg atctgaattt cgaccaccca taatacccat taccctggta gataagtagc 3300
atggcgggtt aatcattaac tacaaggaac ccctagtgat ggagttggcc actccctctc 3360
tgcgcgctcg ctcgctcact gaggccgggc gaccaaaggt cgcccgacgc ccgggctttg 3420
cccgggcggc ctcagtgagc gagcgagcgc gcag 3454
<210> 815
<211> 1602
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 815
atagtctatc caggttgagc atcctgctgg tggttacaag aaactgtttg aaactgtgga 60
ggaactgtcc tcgccgctca cagctcatgt aacaggcagg atccccctct ggctcaccgg 120
cagtctcctt cgatgtgggc caggactctt tgaagttgga tctgagccat tttaccacct 180
gtttgatggg caagccctcc tgcacaagtt tgactttaaa gaaggacatg tcacatacca 240
cagaaggttc atccgcactg atgcttacgt acgggcaatg actgagaaaa ggatcgtcat 300
aacagaattt ggcacctgtg ctttcccaga tccctgcaag aatatatttt ccaggttttt 360
ttcttacttt cgaggagtag aggttactga caattgccct tgttaatgtc tacccagtgg 420
gggaagatta ctacgcttgc acagagacca actttattac aaagattaat ccagagacct 480
tggagacaat taagcaggtt gatctttgca actaagtctc tgtcaatggg gccactgctc 540
acccccacat tgaaaatgat ggaaccgttt acaatattgg taattgcttt ggaaaaaatt 600
tttcaattgc ctacaacatt gtaaagatcc caccactgca agcagacaag gaagatccaa 660
taagcaagtc agagatcgtt gtacaattcc cctgcagtga ccgattcaag ccatcttacg 720
ttcatagttt tggtctgact cccaactata tcgtttttgt ggagacacca gtcaaaatta 780
acctgttcaa gttcctttct tcatggagtc tttggggagc caactacatg gattgttttg 840
agtccaatga aaccatgggg tttggcttca tattgctgac aaaaaaagga aaaagtacct 900
caataataaa tacagaactt ctcctttcaa cctcttccat cacatcaaca cctatgaaga 960
caatgggttt ctgattgtgg atctctgctg ctggaaagga tttgagtttg tttataatta 1020
cttatattta gccaatttac gtgagaactg ggaagaggtg aaaaaaaatg ccagaaaggc 1080
tccccaacct gaagttagga gatatgtact tcctttgaat attgacaagg ctgacacagg 1140
caagaattta gtcagctccc caatacaact gccactgcaa ttctgtgcag tgacgagact 1200
atctggctgg agcctgaagt tctcttttca gggcctcgtc aagcatttga gtttcctcaa 1260
atcaattacc agaagtattg tgggaaacct tacacatatg cgtatggact tggcttgaat 1320
cactttgttc cagataggct ctgtaagctg aatgtcaaaa ctaaagaaac ttgggtttgg 1380
caagagcctg attcataccc atcagaaccc atctttgttt ctcacccaga tgccttggaa 1440
gaagatgatg gtgtagttct gagtgtggtg gtgagcccag gagcaggaca aaagcctgct 1500
tatctcctga ttctgaatgc caaggactta agtgaagttg cccgggctga agtggagatt 1560
aacatccctg tcacctttca tggactgttc aaaaaatctt ga 1602
<210> 816
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 816
cgagtttaat tggtttatag aactcttca 29
<210> 817
<211> 971
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 817
ctgcagaggg ccctgcgtat gagtgcaagt gggttttagg accaggatga ggcggggtgg 60
gggtgcctac ctgacgaccg accccgaccc actggacaag cacccaaccc ccattcccca 120
aattgcgcat cccctatcag agagggggag gggaaacagg atgcggcgag gcgcgtgcgc 180
actgccagct tcagcaccgc ggacagtgcc ttcgcccccg cctggcggcg cgcgccaccg 240
ccgcctcagc actgaaggcg cgctgacgtc actcgccggt cccccgcaaa ctccccttcc 300
cggccacctt ggtcgcgtcc gcgccgccgc cggcccagcc ggaccgcacc acgcgaggcg 360
cgagataggg gggcacgggc gcgaccatct gcgctgcggc gccggcgact cagcgctgcc 420
tcagtctgcg gtgggcagcg gaggagtcgt gtcgtgcctg agagcgcagg gcgcgcctag 480
cccgggctag gtcgactcga ctagggataa cagggtaatt gtttgaatga ggcttcagta 540
ctttacagaa tcgttgcctg cacatcttgg aaacacttgc tgggattact tcttcaggtt 600
aacccaacag aaggctaaag aaggtatatt gctgttgaca gtgagcgacg tctcgatatg 660
gagaacccat gctgtgaagc cacagatggg catgggtttt atatcgagac gctgcctact 720
gcctcggact tcaaggggct actttaggag caattatctt gtttactaaa actgaatacc 780
ttgctatctc tttgatacat ttttacaaag ctgaattaaa atggtataaa ttatcacggg 840
atccgatctt tttccctctg ccaaaaatta tggggacatc atgaagcccc ttgagcatct 900
gacttctggc taataaagga aatttatttt cattgcaata gtgtgttgga attttttgtg 960
tctctcactc g 971
<210> 818
<211> 1543
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 818
ctgcgcgctc gctcgctcac tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtggccaa ctccatcact 120
aggggttcct tgtagttaat gattaacccg ccatgctact tatctaccag ggtaatgggg 180
atcctctaga tccggtcggg cccgcggtac cgtcgagaag cttgatgtgg gcggagcttc 240
gaaggggcgg gcgcccgtgg ggcgggtcct gagtgggggc gggaccgggg ccggcacctg 300
ggtgaggttc tgcagagggc cctgcgtatg agtgcaagtg ggttttagga ccaggatgag 360
gcggggtggg ggtgcctacc tgacgaccga ccccgaccca ctggacaagc acccaacccc 420
cattccccaa attgcgcatc ccctatcaga gagggggagg ggaaacagga tgcggcgagg 480
cgcgtgcgca ctgccagctt cagcaccgcg gacagtgcct tcgcccccgc ctggcggcgc 540
gcgccaccgc cgcctcagca ctgaaggcgc gctgacgtca ctcgccggtc ccccgcaaac 600
tccccttccc ggccaccttg gtcgcgtccg cgccgccgcc ggcccagccg gaccgcacca 660
cgcgaggcgc gagatagggg ggcacgggcg cgaccatctg cgctgcggcg ccggcgactc 720
agcgctgcct cagtctgcgg tgggcagcgg aggagtcgtg tcgtgcctga gagcgcaggg 780
cgcgcctagc ccgggctagg tcgactcgac tagggataac agggtaattg tttgaatgag 840
gcttcagtac tttacagaat cgttgcctgc acatcttgga aacacttgct gggattactt 900
cttcaggtta acccaacaga aggctaaaga aggtatattg ctgttgacag tgagcgacgt 960
ctcgatatgg agaacccatg ctgtgaagcc acagatgggc atgggtttta tatcgagacg 1020
ctgcctactg cctcggactt caaggggcta ctttaggagc aattatcttg tttactaaaa 1080
ctgaatacct tgctatctct ttgatacatt tttacaaagc tgaattaaaa tggtataaat 1140
tatcacggga tccgatcttt ttccctctgc caaaaattat ggggacatca tgaagcccct 1200
tgagcatctg acttctggct aataaaggaa atttattttc attgcaatag tgtgttggaa 1260
ttttttgtgt ctctcactcg gcggccgccc gagtttaatt ggtttataga actcttcaag 1320
ctagcgaagc aattcgttga tctgaatttc gaccacccat aatacccatt accctggtag 1380
ataagtagca tggcgggtta atcattaact acaaggaacc cctagtgatg gagttggcca 1440
ctccctctct gcgcgctcgc tcgctcactg aggccgggcg accaaaggtc gcccgacgcc 1500
cgggctttgc ccgggcggcc tcagtgagcg agcgagcgcg cag 1543
<210> 819
<211> 916
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 819
ctgcagaggg ccctgcgtat gagtgcaagt gggttttagg accaggatga ggcggggtgg 60
gggtgcctac ctgacgaccg accccgaccc actggacaag cacccaaccc ccattcccca 120
aattgcgcat cccctatcag agagggggag gggaaacagg atgcggcgag gcgcgtgcgc 180
actgccagct tcagcaccgc ggacagtgcc ttcgcccccg cctggcggcg cgcgccaccg 240
ccgcctcagc actgaaggcg cgctgacgtc actcgccggt cccccgcaaa ctccccttcc 300
cggccacctt ggtcgcgtcc gcgccgccgc cggcccagcc ggaccgcacc acgcgaggcg 360
cgagataggg gggcacgggc gcgaccatct gcgctgcggc gccggcgact cagcgctgcc 420
tcagtctgcg gtgggcagcg gaggagtcgt gtcgtgcctg agagcgcagg gcgcgcctga 480
attcacgttt ccagaacgtc tgtagctttt ctcctccttc cctccatttt cctcttggtc 540
ttacctttgg cctagtggtt ggtgtagtga taatgtagcg agattttctg cagagcattg 600
cagatggact gggttgcgag gtatgagtaa acagtccata cgcaatgctc cgtggaacgt 660
cacgcagctt tctacagcat gacaagctgc tgaggcttaa atcaggattt tcctgtctct 720
ttctacaaaa tcaaaatgaa aaaagagggc tttttaggca tctccgagat tatgtgctcg 780
aggggatccg atctttttcc ctctgccaaa aattatgggg acatcatgaa gccccttgag 840
catctgactt ctggctaata aaggaaattt attttcattg caatagtgtg ttggaatttt 900
ttgtgtctct cactcg 916
<210> 820
<211> 1488
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 820
ctgcgcgctc gctcgctcac tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtggccaa ctccatcact 120
aggggttcct tgtagttaat gattaacccg ccatgctact tatctaccag ggtaatgggg 180
atcctctaga tccggtcggg cccgcggtac cgtcgagaag cttgatgtgg gcggagcttc 240
gaaggggcgg gcgcccgtgg ggcgggtcct gagtgggggc gggaccgggg ccggcacctg 300
ggtgaggttc tgcagagggc cctgcgtatg agtgcaagtg ggttttagga ccaggatgag 360
gcggggtggg ggtgcctacc tgacgaccga ccccgaccca ctggacaagc acccaacccc 420
cattccccaa attgcgcatc ccctatcaga gagggggagg ggaaacagga tgcggcgagg 480
cgcgtgcgca ctgccagctt cagcaccgcg gacagtgcct tcgcccccgc ctggcggcgc 540
gcgccaccgc cgcctcagca ctgaaggcgc gctgacgtca ctcgccggtc ccccgcaaac 600
tccccttccc ggccaccttg gtcgcgtccg cgccgccgcc ggcccagccg gaccgcacca 660
cgcgaggcgc gagatagggg ggcacgggcg cgaccatctg cgctgcggcg ccggcgactc 720
agcgctgcct cagtctgcgg tgggcagcgg aggagtcgtg tcgtgcctga gagcgcaggg 780
cgcgcctgaa ttcacgtttc cagaacgtct gtagcttttc tcctccttcc ctccattttc 840
ctcttggtct tacctttggc ctagtggttg gtgtagtgat aatgtagcga gattttctgc 900
agagcattgc agatggactg ggttgcgagg tatgagtaaa cagtccatac gcaatgctcc 960
gtggaacgtc acgcagcttt ctacagcatg acaagctgct gaggcttaaa tcaggatttt 1020
cctgtctctt tctacaaaat caaaatgaaa aaagagggct ttttaggcat ctccgagatt 1080
atgtgctcga ggggatccga tctttttccc tctgccaaaa attatgggga catcatgaag 1140
ccccttgagc atctgacttc tggctaataa aggaaattta ttttcattgc aatagtgtgt 1200
tggaattttt tgtgtctctc actcggcggc cgcccgagtt taattggttt atagaactct 1260
tcaagctagc gaagcaattc gttgatctga atttcgacca cccataatac ccattaccct 1320
ggtagataag tagcatggcg ggttaatcat taactacaag gaacccctag tgatggagtt 1380
ggccactccc tctctgcgcg ctcgctcgct cactgaggcc gggcgaccaa aggtcgcccg 1440
acgcccgggc tttgcccggg cggcctcagt gagcgagcga gcgcgcag 1488
<210> 821
<211> 1111
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 821
ctgcagaggg ccctgcgtat gagtgcaagt gggttttagg accaggatga ggcggggtgg 60
gggtgcctac ctgacgaccg accccgaccc actggacaag cacccaaccc ccattcccca 120
aattgcgcat cccctatcag agagggggag gggaaacagg atgcggcgag gcgcgtgcgc 180
actgccagct tcagcaccgc ggacagtgcc ttcgcccccg cctggcggcg cgcgccaccg 240
ccgcctcagc actgaaggcg cgctgacgtc actcgccggt cccccgcaaa ctccccttcc 300
cggccacctt ggtcgcgtcc gcgccgccgc cggcccagcc ggaccgcacc acgcgaggcg 360
cgagataggg gggcacgggc gcgaccatct gcgctgcggc gccggcgact cagcgctgcc 420
tcagtctgcg gtgggcagcg gaggagtcgt gtcgtgcctg agagcgcagg gcgcgcctga 480
attctctgcc gcggaaaggg gagaagtgtg ggctcctccg agtcgggggc ggactgggac 540
agcacagtcg gctgagcgca gcgcccccgc cctgcccgcc acgcggcgaa gacgcctgag 600
cgttcgcgcc cctcgggcga ggaccccacg caagcccgag ccggtcccga ccctggcccc 660
gacgctcgcc gcccgcccca gccctgaggg cccctcgacg tttatctaca acactctgat 720
ttaatgtcta tacaatcaga gcattgcaga tggactgcga gaggcgcctc cgccgctcct 780
ttctcatgga aatggcccgc gagcccgtcc ggcccagcgc ccctcccgcg ggaggaaggc 840
gagcccggcc cccggcggcc attcgcgccg cggacaaatc cggcgaacaa tgcgcccgcc 900
cagagtgcgg cccagctgcc gggccgggga tctggccgcg ggacacaaag gggcccgcac 960
gcctctggcg tctcgagggg atccgatctt tttccctctg ccaaaaatta tggggacatc 1020
atgaagcccc ttgagcatct gacttctggc taataaagga aatttatttt cattgcaata 1080
gtgtgttgga attttttgtg tctctcactc g 1111
<210> 822
<211> 1683
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 822
ctgcgcgctc gctcgctcac tgaggccgcc cgggcaaagc ccgggcgtcg ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtggccaa ctccatcact 120
aggggttcct tgtagttaat gattaacccg ccatgctact tatctaccag ggtaatgggg 180
atcctctaga tccggtcggg cccgcggtac cgtcgagaag cttgatgtgg gcggagcttc 240
gaaggggcgg gcgcccgtgg ggcgggtcct gagtgggggc gggaccgggg ccggcacctg 300
ggtgaggttc tgcagagggc cctgcgtatg agtgcaagtg ggttttagga ccaggatgag 360
gcggggtggg ggtgcctacc tgacgaccga ccccgaccca ctggacaagc acccaacccc 420
cattccccaa attgcgcatc ccctatcaga gagggggagg ggaaacagga tgcggcgagg 480
cgcgtgcgca ctgccagctt cagcaccgcg gacagtgcct tcgcccccgc ctggcggcgc 540
gcgccaccgc cgcctcagca ctgaaggcgc gctgacgtca ctcgccggtc ccccgcaaac 600
tccccttccc ggccaccttg gtcgcgtccg cgccgccgcc ggcccagccg gaccgcacca 660
cgcgaggcgc gagatagggg ggcacgggcg cgaccatctg cgctgcggcg ccggcgactc 720
agcgctgcct cagtctgcgg tgggcagcgg aggagtcgtg tcgtgcctga gagcgcaggg 780
cgcgcctgaa ttctctgccg cggaaagggg agaagtgtgg gctcctccga gtcgggggcg 840
gactgggaca gcacagtcgg ctgagcgcag cgcccccgcc ctgcccgcca cgcggcgaag 900
acgcctgagc gttcgcgccc ctcgggcgag gaccccacgc aagcccgagc cggtcccgac 960
cctggccccg acgctcgccg cccgccccag ccctgagggc ccctcgacgt ttatctacaa 1020
cactctgatt taatgtctat acaatcagag cattgcagat ggactgcgag aggcgcctcc 1080
gccgctcctt tctcatggaa atggcccgcg agcccgtccg gcccagcgcc cctcccgcgg 1140
gaggaaggcg agcccggccc ccggcggcca ttcgcgccgc ggacaaatcc ggcgaacaat 1200
gcgcccgccc agagtgcggc ccagctgccg ggccggggat ctggccgcgg gacacaaagg 1260
ggcccgcacg cctctggcgt ctcgagggga tccgatcttt ttccctctgc caaaaattat 1320
ggggacatca tgaagcccct tgagcatctg acttctggct aataaaggaa atttattttc 1380
attgcaatag tgtgttggaa ttttttgtgt ctctcactcg gcggccgccc gagtttaatt 1440
ggtttataga actcttcaag ctagcgaagc aattcgttga tctgaatttc gaccacccat 1500
aatacccatt accctggtag ataagtagca tggcgggtta atcattaact acaaggaacc 1560
cctagtgatg gagttggcca ctccctctct gcgcgctcgc tcgctcactg aggccgggcg 1620
accaaaggtc gcccgacgcc cgggctttgc ccgggcggcc tcagtgagcg agcgagcgcg 1680
cag 1683
<210> 823
<211> 1602
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 823
atagtctatc caggttgagc atcctgctgg tggttacaag aaactgtttg aaactgtgga 60
ggaactgtcc tcgccgctca cagctcatgt aacaggcagg atccccctct ggctcaccgg 120
cagtctcctt cgatgtgggc caggactctt tgaagttgga tctgagccat tttaccacct 180
gtttgatggg caagccctcc tgcacaagtt tgactttaaa gaaggacatg tcacatacca 240
cagaaggttc atccgcactg atgcttacgt acgggcaatg actgagaaaa ggatcgtcat 300
aacagaattt ggcacctgtg ctttcccaga tccctgcaag aatatatttt ccaggttttt 360
ttcttacttt cgaggagtag aggttactga caattgccct tgttaatgtc tacccagtgg 420
gggaagatta ctacgcttgc acagagacca actttattac aaagattaat ccagagacct 480
tggagacaat taagcaggtt gatctttgca actaagtctc tgtcaatggg gccactgctc 540
acccccacat tgaaaatgat ggaaccgttt acaatattgg taattgcttt ggaaaaaatt 600
tttcaattgc ctacaacatt gtaaagatcc caccactgca agcagacaag gaagatccaa 660
taagcaagtc agagatcgtt gtacaattcc cctgcagtga ccgattcaag ccatcttacg 720
ttcatagttt tggtctgact cccaactata tcgtttttgt ggagacacca gtcaaaatta 780
acctgttcaa gttcctttct tcatggagtc tttggggagc caactacatg gattgttttg 840
agtccaatga aaccatgggg tttggcttca tattgctgac aaaaaaagga aaaagtacct 900
caataataaa tacagaactt ctcctttcaa cctcttccat cacatcaaca cctatgaaga 960
caatgggttt ctgattgtgg atctctgctg ctggaaagga tttgagtttg tttataatta 1020
cttatattta gccaatttac gtgagaactg ggaagaggtg aaaaaaaatg ccagaaaggc 1080
tccccaacct gaagttagga gatatgtact tcctttgaat attgacaagg ctgacacagg 1140
caagaattta gtcagctccc caatacaact gccactgcaa ttctgtgcag tgacgagact 1200
atctggctgg agcctgaagt tctcttttca gggcctcgtc aagcatttga gtttcctcaa 1260
atcaattacc agaagtattg tgggaaacct tacacatatg cgtatggact tggcttgaat 1320
cactttgttc cagataggct ctgtaagctg aatgtcaaaa ctaaagaaac ttgggtttgg 1380
caagagcctg attcataccc atcagaaccc atctttgttt ctcacccaga tgccttggaa 1440
gaagatgatg gtgtagttct gagtgtggtg gtgagcccag gagcaggaca aaagcctgct 1500
tatctcctga ttctgaatgc caaggactta agtgaagttg cccgggctga agtggagatt 1560
aacatccctg tcacctttca tggactgttc aaaaaatctt ga 1602
<210> 824
<211> 837
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 824
gtgagcggcg gatgcccttc tcctctggct gtaattagtc ttggtttact tgtttctttt 60
ctgtggctgt tgaaagcctt gaggggctct ggagggccct ttgtgtgggg gagtgcttgg 120
gggtgttgtt gtgtgtgtgt tggggagtct ttgtgctctt ctgcctgtgc tgtgagtctg 180
tggttgttgg gctttgtgtc tctcagtgtg ttaggggagt tgctggggtg tgcccttgtg 240
tgggggggct gtaggggaac aaaggctgtt gtgggtgtgt gttggggggg tgagcagggg 300
gtgtgggttt tgttggctgc aaccccccct gcacccccct ccctagttgc tgagcatgcc 360
tgctttggtg tgggctctta tgggttggtt gggcttcttg ctggtggggg tggtgcaggt 420
gggggtgctg gtgggtgggc tccttggctg ggagggcttg gggaggggtt gtgcccctga 480
gtctgtgctg ttaggttgta gctcagccat tgcctttttt gtagagggtc agggacttcc 540
tttgtcccaa atctgtgtga gctaaatctg ggaggtctct caccccctct agtggttggg 600
taagtgtgtg tctgcaggaa ggaaagggtg ggagggcctt ttgtttcttc tcttcccctt 660
ctccctctcc agccttgggc tgtcttgggg gatgctgcct ttggggggat gggcagggtg 720
ggtttgcttc tggttgtgac tgtgctctag agcctctgct aacctgttct gccttcttct 780
ttttcctaca gctcctgggc aattgctggt tattgtgctg tctctatttt ggcaaag 837
Claims (140)
- Grik2 mRNA의 단일 가닥 영역 내에 특이적으로 혼성화하는 800개 이내 뉴클레오티드의 길이를 갖는 단리된 폴리뉴클레오티드로서, 여기서 상기 혼성화된 폴리뉴클레오티드는 18 kcal/mol보다 작은 표적 개방 에너지를 갖고, 그리고
(a) 상기 폴리뉴클레오티드는 서열 번호: 772-774 중에서 하나의 핵산 서열을 포함하지 않거나;
(b) 상기 폴리뉴클레오티드는 서열 번호: 68 또는 서열 번호: 68 및 서열 번호: 649의 핵산 서열을 포함하거나; 또는
(c) 상기 폴리뉴클레오티드는 5.53 kcal/mol 및 5.55 kcal/mol 사이에 있는 총 개방 에너지를 갖지 않는, 단리된 폴리뉴클레오티드. - Grik2 mRNA의 단일 가닥 영역 내에 특이적으로 혼성화하는 23개 이내 뉴클레오티드의 길이를 갖는 단리된 RNA 폴리뉴클레오티드로서, 여기서 상기 혼성화된 폴리뉴클레오티드는 18 kcal/mol보다 작은 총 개방 에너지를 갖고, 여기서 상기 폴리뉴클레오티드는 서열 번호: 772-774 중에서 하나의 핵산 서열을 포함하지 않는, 단리된 RNA 폴리뉴클레오티드.
- 청구항 제1항 또는 제2항에 있어서, 폴리뉴클레오티드는 5.4 kcal/mol보다 크거나 또는 5.4 kcal/mol보다 작은 총 개방 에너지를 갖는, 폴리뉴클레오티드.
- 청구항 제1항 내지 제3항 중 어느 한 항에 있어서, 혼성화된 폴리뉴클레오티드는 -35 kcal/mol보다 큰 이중나선 형성의 에너지를 갖는, 폴리뉴클레오티드.
- 청구항 제1항 내지 제4항 중 어느 한 항에 있어서, 혼성화된 폴리뉴클레오티드는 -24 kcal/mol보다 큰 총 결합 에너지를 갖는, 폴리뉴클레오티드.
- 청구항 제1항 내지 제5항 중 어느 한 항에 있어서, 혼성화된 폴리뉴클레오티드는 50%보다 적은 GC 함량을 갖는, 폴리뉴클레오티드.
- 청구항 제1항 내지 제6항 중 어느 한 항에 있어서, Grik2 mRNA의 단일 가닥 영역은 루프 영역 1-14로 구성된 군에서 선택되는, 폴리뉴클레오티드.
- 청구항 제1항 내지 제7항 중 어느 한 항에 있어서, 폴리뉴클레오티드는
(a) Grik2 mRNA의 루프 1 영역;
(b) Grik2 mRNA의 루프 2 영역;
(c) Grik2 mRNA의 루프 3 영역;
(d) Grik2 mRNA의 루프 4 영역;
(e) Grik2 mRNA의 루프 5 영역;
(f) Grik2 mRNA의 루프 6 영역;
(g) Grik2 mRNA의 루프 7 영역;
(h) Grik2 mRNA의 루프 8 영역;
(i) Grik2 mRNA의 루프 9 영역;
(j) Grik2 mRNA의 루프 10 영역;
(k) Grik2 mRNA의 루프 11 영역;
(l) Grik2 mRNA의 루프 12 영역;
(m) Grik2 mRNA의 루프 13 영역;
(n) Grik2 mRNA의 루프 14 영역 내에 특이적으로 혼성화하는, 폴리뉴클레오티드. - 청구항 제7항 또는 청구항 제8항에 있어서,
(a) 루프 1 영역은 서열 번호: 145의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(b) 루프 2 영역은 서열 번호: 146의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(c) 루프 3 영역은 서열 번호: 147의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(d) 루프 4 영역은 서열 번호: 148의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(e) 루프 5 영역은 서열 번호: 149의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(f) 루프 6 영역은 서열 번호: 150의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(g) 루프 7 영역은 서열 번호: 151의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(h) 루프 8 영역은 서열 번호: 152의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(i) 루프 9 영역은 서열 번호: 153의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(j) 루프 10 영역은 서열 번호: 154의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(k) 루프 11 영역은 서열 번호: 155의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(l) 루프 12 영역은 서열 번호: 156의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(m) 루프 13 영역은 서열 번호: 157의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고; 및/또는
(n) 루프 14 영역은 서열 번호: 158의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되는, 폴리뉴클레오티드. - 청구항 제9항에 있어서, 서열 동일성은 서열 번호: 145-158 중에서 하나의 적어도 15개의 연속 뉴클레오티드에 걸쳐 결정되는, 폴리뉴클레오티드.
- 청구항 제10항에 있어서, 서열 동일성은 서열 번호: 145-158 중에서 하나의 적어도 30개의 연속 뉴클레오티드에 걸쳐 결정되는, 폴리뉴클레오티드.
- 청구항 제11항에 있어서, 서열 동일성은 서열 번호: 145-158 중에서 하나의 적어도 60개의 연속 뉴클레오티드에 걸쳐 결정되는, 폴리뉴클레오티드.
- 청구항 제12항에 있어서, 서열 동일성은 서열 번호: 145-158 중에서 하나의 전장에 걸쳐 결정되는, 폴리뉴클레오티드.
- 청구항 제1항 내지 제13항 중 어느 한 항에 있어서, 폴리뉴클레오티드는
(a) 서열 번호: 1의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(b) 서열 번호: 4의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(c) (i) 서열 번호: 5의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 또는
(ii) 서열 번호: 6의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(d) 서열 번호: 7의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(e) 서열 번호: 96의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(f) (i) 서열 번호: 8의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 98의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 또는
(iii) 서열 번호: 99의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(g) 서열 번호: 9의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(h) 서열 번호: 63의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(i) 서열 번호: 10의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 또는
(j) 서열 번호: 11의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함하는, 뉴클레오티드. - 청구항 제14항에 있어서, 서열 동일성은 서열 번호: 1, 4-11, 63, 96, 98 또는 99 중에서 하나의 적어도 10개의 연속 뉴클레오티드에 걸쳐 결정되는, 폴리뉴클레오티드.
- 청구항 제15항에 있어서, 서열 동일성은 서열 번호: 1, 4-11, 63, 96, 98 또는 99 중에서 하나의 적어도 15개의 연속 뉴클레오티드에 걸쳐 결정되는, 폴리뉴클레오티드.
- 청구항 제16항에 있어서, 서열 동일성은 서열 번호: 1, 4-11, 63, 96, 98 또는 99 중에서 하나의 적어도 20개의 연속 뉴클레오티드에 걸쳐 결정되는, 폴리뉴클레오티드.
- 청구항 제17항에 있어서, 서열 동일성은 서열 번호: 1, 4-11, 63, 96, 98 또는 99 중에서 하나의 전장에 걸쳐 결정되는, 폴리뉴클레오티드.
- 청구항 제1항 내지 제18항 중 어느 한 항에 있어서, 폴리뉴클레오티드는 상기 폴리뉴클레오티드 및 Grik2 mRNA의 단일 가닥 영역에 의해 형성된 이중나선 구조를 포함하고, 여기서 상기 이중나선 구조는 상기 폴리뉴클레오티드의 뉴클레오티드 및 Grik2 mRNA의 단일 가닥 영역의 뉴클레오티드 사이에 적어도 하나의 부정합을 포함하는, 폴리뉴클레오티드.
- 청구항 제19항에 있어서, Grik2 mRNA의 단일 가닥 영역은 루프 영역 1-14로 구성된 군에서 선택되는, 폴리뉴클레오티드.
- 청구항 제1항 내지 제20항 중 어느 한 항에 있어서, 총 개방 에너지, 이중나선 형성의 에너지, 총 결합 에너지 및/또는 GC 함량은 23개 내지 79개 뉴클레오티드에 걸쳐 계산되는, 폴리뉴클레오티드.
- 청구항 제1항 내지 제7항 중 어느 한 항에 있어서, Grik2 mRNA의 단일 가닥 영역은 비대합 영역 1-5로 구성된 군에서 선택되는, 폴리뉴클레오티드.
- 청구항 제22항에 있어서, 폴리뉴클레오티드는
(a) Grik2 mRNA의 비대합 영역 1;
(b) Grik2 mRNA의 비대합 영역 2;
(c) Grik2 mRNA의 비대합 영역 3;
(d) Grik2 mRNA의 비대합 영역 4; 또는
(e) Grik2 mRNA의 비대합 영역 5 내에 특이적으로 혼성화하는, 폴리뉴클레오티드. - 청구항 제22항 또는 제23항에 있어서,
(a) 비대합 영역 1은 서열 번호: 159의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(b) 비대합 영역 2는 서열 번호: 160의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(c) 비대합 영역 3은 서열 번호: 161의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(d) 비대합 영역 4는 서열 번호: 162의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고; 및/또는
(e) 비대합 영역 5는 서열 번호: 163의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되는, 폴리뉴클레오티드. - 청구항 제24항에 있어서, 서열 동일성은 서열 번호: 159-163 중에서 하나의 적어도 15개의 연속 뉴클레오티드에 걸쳐 결정되는, 폴리뉴클레오티드.
- 청구항 제25항에 있어서, 서열 동일성은 서열 번호: 159-163 중에서 하나의 적어도 30개의 연속 뉴클레오티드에 걸쳐 결정되는, 폴리뉴클레오티드.
- 청구항 제26항에 있어서, 서열 동일성은 서열 번호: 159-163 중에서 하나의 적어도 60개의 연속 뉴클레오티드에 걸쳐 결정되는, 폴리뉴클레오티드.
- 청구항 제27항에 있어서, 서열 동일성은 서열 번호: 159-163 중에서 하나의 전장에 걸쳐 결정되는, 폴리뉴클레오티드.
- 청구항 제22항 내지 제28항 중 어느 한 항에 있어서, 폴리뉴클레오티드는
(a) (i) 서열 번호: 13의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 14의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iii) 서열 번호: 72의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 또는
(iv) 서열 번호: 73의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(b) 서열 번호: 15의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 또는
(c) 서열 번호: 16의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함하는, 폴리뉴클레오티드. - 청구항 제29항에 있어서, 서열 동일성은 서열 번호: 13-16, 72 또는 73 중에서 하나의 적어도 10개의 연속 뉴클레오티드에 걸쳐 결정되는, 폴리뉴클레오티드.
- 청구항 제30항에 있어서, 서열 동일성은 서열 번호: 13-16, 72 또는 73 중에서 하나의 적어도 15개의 연속 뉴클레오티드에 걸쳐 결정되는, 폴리뉴클레오티드.
- 청구항 제31항에 있어서, 서열 동일성은 서열 번호: 13-16, 72 또는 73 중에서 하나의 적어도 20개의 연속 뉴클레오티드에 걸쳐 결정되는, 폴리뉴클레오티드.
- 청구항 제32항에 있어서, 서열 동일성은 서열 번호: 13-16, 72 또는 73 중에서 하나의 전장에 걸쳐 결정되는, 폴리뉴클레오티드.
- 청구항 제29항 내지 제33항 중 어느 한 항에 있어서, 서열 동일성은 서열 번호: 13-16, 72 또는 73 중에서 하나의 30개 이내의 연속 뉴클레오티드에 걸쳐 결정되는, 폴리뉴클레오티드.
- 청구항 제34항에 있어서, 서열 동일성은 서열 번호: 13-16, 72 또는 73 중에서 하나의 25개 이내의 연속 뉴클레오티드에 걸쳐 결정되는, 폴리뉴클레오티드.
- 청구항 제22항 내지 제35항 중 어느 한 항에 있어서, 폴리뉴클레오티드는 상기 폴리뉴클레오티드 및 Grik2 mRNA의 단일 가닥 영역에 의해 형성된 이중나선 구조를 포함하고, 여기서 상기 이중나선 구조는 상기 폴리뉴클레오티드의 뉴클레오티드 및 Grik2 mRNA의 단일 가닥 영역의 뉴클레오티드 사이에 적어도 하나의 부정합을 포함하는, 폴리뉴클레오티드.
- 청구항 제22항 내지 제36항 중 어느 한 항에 있어서, 평균 위치 엔트로피가 23개 내지 79개 뉴클레오티드에 걸쳐 계산되는, 폴리뉴클레오티드.
- 청구항 제1항 내지 제37항 중 어느 한 항에 있어서, 폴리뉴클레오티드는 Grik2 mRNA의 코딩 서열에 혼성화하는, 폴리뉴클레오티드.
- 청구항 제38항에 있어서, 폴리뉴클레오티드는
(a) Grik2 mRNA의 엑손 1 내에 영역;
(b) Grik2 mRNA의 엑손 2 내에 영역;
(c) Grik2 mRNA의 엑손 3 내에 영역;
(d) Grik2 mRNA의 엑손 4 내에 영역;
(e) Grik2 mRNA의 엑손 5 내에 영역;
(f) Grik2 mRNA의 엑손 6 내에 영역;
(g) Grik2 mRNA의 엑손 7 내에 영역;
(h) Grik2 mRNA의 엑손 8 내에 영역;
(i) Grik2 mRNA의 엑손 9 내에 영역;
(j) Grik2 mRNA의 엑손 10 내에 영역;
(k) Grik2 mRNA의 엑손 11 내에 영역;
(l) Grik2 mRNA의 엑손 12 내에 영역;
(m) Grik2 mRNA의 엑손 13 내에 영역;
(n) Grik2 mRNA의 엑손 14 내에 영역;
(o) Grik2 mRNA의 엑손 15 내에 영역; 및/또는
(p) Grik2 mRNA의 엑손 16 내에 영역에 혼성화하는, 폴리뉴클레오티드. - 청구항 제39항에 있어서,
(a) Grik2 mRNA의 엑손 1은 서열 번호: 129의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(b) Grik2 mRNA의 엑손 2는 서열 번호: 130의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(c) Grik2 mRNA의 엑손 3은 서열 번호: 131의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(d) Grik2 mRNA의 엑손 4는 서열 번호: 132의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(e) Grik2 mRNA의 엑손 5는 서열 번호: 133의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(f) Grik2 mRNA의 엑손 6은 서열 번호: 134의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(g) Grik2 mRNA의 엑손 7은 서열 번호: 135의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(h) Grik2 mRNA의 엑손 8은 서열 번호: 136의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(i) Grik2 mRNA의 엑손 9는 서열 번호: 137의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(j) Grik2 mRNA의 엑손 10은 서열 번호: 138의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(k) Grik2 mRNA의 엑손 11은 서열 번호: 139의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(l) Grik2 mRNA의 엑손 12는 서열 번호: 140의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(m) Grik2 mRNA의 엑손 13은 서열 번호: 141의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(n) Grik2 mRNA의 엑손 14는 서열 번호: 142의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고;
(o) Grik2 mRNA의 엑손 15는 서열 번호: 143의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되고; 및/또는
(p) Grik2 mRNA의 엑손 16은 서열 번호: 144의 적어도 10개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열에 의해 인코딩되는, 폴리뉴클레오티드. - 청구항 제40항에 있어서, 폴리뉴클레오티드는
(a) 서열 번호: 1의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(b) (i) 서열 번호: 2의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 3의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iii) 서열 번호: 30의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iv) 서열 번호: 31의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(v) 서열 번호: 36의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(vi) 서열 번호: 40의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(vii) 서열 번호: 59의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(viii) 서열 번호: 76의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ix) 서열 번호: 80의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(x) 서열 번호: 81의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(xi) 서열 번호: 92의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 및/또는
(xii) 서열 번호: 93의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(c) (i) 서열 번호: 40의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 60의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iii) 서열 번호: 68의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iv) 서열 번호: 70의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 및/또는
(v) 서열 번호: 86의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(d) (i) 서열 번호: 68의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 69의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 및/또는
(iii) 서열 번호: 70의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(e) (i) 서열 번호: 4의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 5의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iii) 서열 번호: 6의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(vi) 서열 번호: 56의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(v) 서열 번호: 57의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(vi) 서열 번호: 58의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(vii) 서열 번호: 91의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(viii) 서열 번호: 94의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 및/또는
(ix) 서열 번호: 95의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(f) (i) 서열 번호: 20의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 37의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iii) 서열 번호: 38의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iv) 서열 번호: 44의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(v) 서열 번호: 46의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(g) 서열 번호: 12의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(h) (i) 서열 번호: 7의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 8의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iii) 서열 번호: 96의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iv) 서열 번호: 98의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 및/또는
(v) 서열 번호: 99의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(i) (i) 서열 번호: 22의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 39의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iii) 서열 번호: 62의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(vi) 서열 번호: 74의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(v) 서열 번호: 75의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(vi) 서열 번호: 87의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(vii) 서열 번호: 88의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(viii) 서열 번호: 89의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 및/또는
(ix) 서열 번호: 90의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(j) (i) 서열 번호: 82의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 83의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iii) 서열 번호: 84의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 및/또는
(iv) 서열 번호: 85의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(k) (i) 서열 번호: 13의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 14의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iii) 서열 번호: 72의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 및/또는
(iv) 서열 번호: 73의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(l) (i) 서열 번호: 34의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 35의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iii) 서열 번호: 77의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iv) 서열 번호: 78의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 및/또는
(v) 서열 번호: 79의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(m) 서열 번호: 51의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
및/또는
(n) (i) 서열 번호: 9의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ii) 서열 번호: 10의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iii) 서열 번호: 11의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(iv) 서열 번호: 15의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(v) 서열 번호: 16의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(vi) 서열 번호: 17의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(vii) 서열 번호: 18의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(viii) 서열 번호: 27의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(ix) 서열 번호: 32의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(x) 서열 번호: 33의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(xi) 서열 번호: 41의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(xii) 서열 번호: 49의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(xiii) 서열 번호: 50의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(xiv) 서열 번호: 52의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(xv) 서열 번호: 53의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열;
(xvi) 서열 번호: 61의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열; 및/또는
(xvii) 서열 번호: 63의 적어도 5개의 연속 뉴클레오티드에 걸쳐 적어도 85%, 90%, 92%, 95%, 97%, 99% 또는 100% 서열 동일성을 갖는 핵산 서열을 포함하는, 폴리뉴클레오티드. - 청구항 제41항에 있어서, 서열 동일성은 서열 번호: 1-12, 13-18, 20, 22, 27, 30-41, 44, 46, 49-53, 56-63, 68-70, 72-92, 또는 94-99 중에서 하나의 적어도 10개의 연속 뉴클레오티드에 걸쳐 결정되는, 폴리뉴클레오티드.
- 청구항 제42항에 있어서, 서열 동일성은 서열 번호: 1-12, 13-18, 20, 22, 27, 30-41, 44, 46, 49-53, 56-63, 68-70, 72-92, 또는 94-99 중에서 하나의 적어도 15개의 연속 뉴클레오티드에 걸쳐 결정되는, 폴리뉴클레오티드.
- 청구항 제43항에 있어서, 서열 동일성은 서열 번호: 1-12, 13-18, 20, 22, 27, 30-41, 44, 46, 49-53, 56-63, 68-70, 72-92, 또는 94-99 중에서 하나의 적어도 20개의 연속 뉴클레오티드에 걸쳐 결정되는, 폴리뉴클레오티드.
- 청구항 제44항에 있어서, 서열 동일성은 서열 번호: 1-12, 13-18, 20, 22, 27, 30-41, 44, 46, 49-53, 56-63, 68-70, 72-92, 또는 94-99 중에서 하나의 전장에 걸쳐 결정되는, 폴리뉴클레오티드.
- 청구항 제1항에 있어서, 폴리뉴클레오티드는 서열 번호: 68 및 서열 번호: 649의 핵산 서열을 포함하는, 폴리뉴클레오티드.
- 청구항 제1항 내지 제46항 중 어느 한 항에 있어서, 폴리뉴클레오티드는 Grik2 mRNA의 비코딩 서열에 혼성화하는, 폴리뉴클레오티드.
- 청구항 제47항에 있어서, 비코딩 서열은 Grik2 mRNA의 5' 비번역 영역 (UTR)을 포함하는, 폴리뉴클레오티드.
- 청구항 제48항에 있어서, 5' UTR은 서열 번호: 126의 핵산 서열에 적어도 85% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 인코딩되는, 폴리뉴클레오티드.
- 청구항 제47항에 있어서, 비코딩 서열은 Grik2 mRNA의 3' UTR을 포함하는, 폴리뉴클레오티드.
- 청구항 제50항에 있어서, 3' UTR은 서열 번호: 127의 핵산 서열에 적어도 85% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 인코딩되는, 폴리뉴클레오티드.
- 청구항 제1항 내지 제51항 중 어느 한 항에 있어서, 폴리뉴클레오티드는 서열 번호: 115-681의 핵산 서열 중에서 하나에 혼성화하는, 폴리뉴클레오티드.
- 청구항 제1항 내지 제52항 중 어느 한 항에 있어서, 폴리뉴클레오티드는 서열 번호: 1-100 중에서 하나의 핵산 서열에 적어도 85% 서열 동일성을 갖는, 폴리뉴클레오티드.
- 청구항 제1항 내지 제53항 중 어느 한 항에 있어서, 폴리뉴클레오티드는 안티센스 올리고뉴클레오티드 (ASO)인, 폴리뉴클레오티드.
- 청구항 제54항에 있어서, ASO는 짧은 간섭 RNA (siRNA), 짧은 헤어핀 RNA (shRNA), 마이크로RNA (miRNA), 또는 짧은 헤어핀 적응 miRNA (shmiRNA)인, 폴리뉴클레오티드.
- 청구항 제1항 내지 제55항 중 어느 한 항에 있어서, 폴리뉴클레오티드는 19개 내지 21개 뉴클레오티드 사이인, 폴리뉴클레오티드.
- 청구항 제56항에 있어서, 폴리뉴클레오티드는 19개 뉴클레오티드인, 폴리뉴클레오티드.
- 청구항 제57항에 있어서, 폴리뉴클레오티드는 20개 뉴클레오티드인, 폴리뉴클레오티드.
- 청구항 제58항에 있어서, 폴리뉴클레오티드는 21개 뉴클레오티드인, 폴리뉴클레오티드.
- 청구항 제1항 내지 제59항 중 어느 한 항에 있어서, Grik2 mRNA는 서열 번호: 115, 서열 번호: 116, 서열 번호: 117, 서열 번호: 118, 서열 번호: 119, 서열 번호: 120, 서열 번호: 121, 서열 번호: 122, 서열 번호: 123, 또는 서열 번호: 124의 핵산 서열에 의해 인코딩되는, 폴리뉴클레오티드.
- 청구항 제1항 내지 제60항 중 어느 한 항에 있어서, 폴리뉴클레오티드는 세포에서 Gluk2 단백질의 수준을 감소시킬 수 있는, 폴리뉴클레오티드.
- 청구항 제61항에 있어서, 폴리뉴클레오티드는 세포에서 GluK2 단백질의 수준을 적어도 10%, 적어도 15%, 적어도 20%, 적어도 25%, 적어도 30%, 적어도 35%, 적어도 40%, 적어도 45%, 적어도 50%, 적어도 55%, 적어도 60%, 적어도 65%, 적어도 70%, 또는 적어도 75% 감소시키는, 폴리뉴클레오티드.
- 청구항 제61항 또는 제62항에 있어서, 세포는 뉴런인, 폴리뉴클레오티드.
- 청구항 제63항에 있어서, 뉴런은 해마 뉴런인, 폴리뉴클레오티드.
- 청구항 제64항에 있어서, 해마 뉴런은 치상 과립 세포 (DGC)인, 폴리뉴클레오티드.
- 청구항 제1항 내지 제65항 중 어느 한 항의 폴리뉴클레오티드를 포함하는 벡터.
- 청구항 제66항에 있어서, 벡터는 복제 결함성인, 벡터.
- 청구항 제66항 또는 제67항에 있어서, 벡터는 포유류, 세균 또는 바이러스 벡터인, 벡터.
- 청구항 제66항 내지 제68항 중 어느 한 항에 있어서, 벡터는 발현 벡터인, 벡터.
- 청구항 제68항 또는 청구항 제69항에 있어서, 바이러스 벡터는 아데노 관련 바이러스 (AAV), 레트로바이러스, 아데노바이러스, 파보바이러스, 코로나바이러스, 음성 가닥 RNA 바이러스, 오르토믹소바이러스, 랍도바이러스, 파라믹소바이러스, 양성 가닥 RNA 바이러스, 피코르나바이러스, 알파바이러스, 이중 가닥 DNA 바이러스, 헤르페스바이러스, 엡스타인 바르 바이러스, 시토메갈로바이러스, 계두바이러스, 그리고 카나리아두바이러스로 구성된 군에서 선택되는, 벡터.
- 청구항 제70항에 있어서, 벡터는 AAV 벡터인, 벡터.
- 청구항 제71항에 있어서, AAV 벡터는 AAV9 또는 AAVrh10 벡터인, 벡터.
- 청구항 제66항 내지 제72항 중 어느 한 항에 있어서, 벡터는 표 9에서 선택되는 발현 카세트를 포함하는, 벡터.
- 다음을 포함하는 뉴클레오티드 서열을 포함하는 발현 카세트:
(a) 스템 루프 서열로서, 5'에서 3'로:
(i) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나에 적어도 85% 서열 동일성을 갖는 가이드 뉴클레오티드 서열을 포함하는 5' 스템 루프 팔;
(ii) 루프 영역으로서, 여기서 상기 루프 영역은 마이크로RNA 루프 서열을 포함하고;
(iii) 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 승객 뉴클레오티드 서열을 포함하는 3' 스템 루프 팔을 포함하는 스템 루프 서열,
(b) 상기 가이드 서열의 5'에 위치된 첫 번째 측부 영역; 및
(c) 상기 승객 서열의 3'에 위치된 두 번째 측부 영역. - 청구항 제74항에 있어서, 발현 카세트는 표 9에서 설명된 구조 중에서 하나를 포함하는, 발현 카세트.
- 다음을 포함하는 뉴클레오티드 서열을 포함하는 발현 카세트:
(a) 스템 루프 서열로서, 5'에서 3'로:
(i) 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 승객 뉴클레오티드 서열을 포함하는 5' 스템 루프 팔;
(ii) 루프 영역으로서, 여기서 상기 루프 영역은 마이크로RNA 루프 서열을 포함하고;
(iii) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나에 적어도 85% 서열 동일성을 갖는 가이드 뉴클레오티드 서열을 포함하는 3' 스템 루프 팔을 포함하는 스템 루프 서열;
(b) 상기 가이드 서열의 5'에 위치된 첫 번째 측부 영역; 및
(c) 상기 승객 서열의 3'에 위치된 두 번째 측부 영역. - 청구항 제76항에 있어서, 발현 카세트는 표 9에서 설명된 구조 중에서 하나를 포함하는, 발현 카세트.
- 청구항 제74항 내지 제77항 중 어느 한 항에 있어서, 첫 번째 측부 영역은 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나의 핵산 서열에 적어도 85% 서열 동일성을 갖는 폴리뉴클레오티드를 포함하는, 발현 카세트.
- 청구항 제74항 내지 제78항 중 어느 한 항에 있어서, 두 번째 측부 영역은 서열 번호: 753, 755, 757, 760, 763, 766 또는 769 중에서 하나의 핵산 서열에 적어도 85% 서열 동일성을 갖는 폴리뉴클레오티드를 포함하는, 발현 카세트.
- 청구항 제74항 내지 제79항 중 어느 한 항에 있어서, 첫 번째 측부 영역은 5' 스페이서 서열 및 5' 측부 서열을 포함하는, 발현 카세트.
- 청구항 제74항 내지 제80항 중 어느 한 항에 있어서, 두 번째 측부 영역은 3' 스페이서 서열 및 3' 측부 서열을 포함하는, 발현 카세트.
- 청구항 제74항 내지 제81항 중 어느 한 항에 있어서, 마이크로RNA 루프 서열은 miR-30, miR-155, miR-218-1, 또는 miR-124-3 서열인, 발현 카세트.
- 청구항 제82항에 있어서, 마이크로RNA 루프 서열은 서열 번호: 758, 761, 764, 767 또는 770 중에서 하나의 핵산 서열에 적어도 85% 서열 동일성을 갖는 폴리뉴클레오티드를 포함하는, 발현 카세트.
- 청구항 제74항 내지 제83항 중 어느 한 항에 있어서, 발현 카세트는 U6 프로모터, H1 프로모터, 7SK 프로모터, 아포지질단백질 E-인간 알파 1-항트립신 프로모터, CAG 프로모터, CBA 프로모터, CK8 프로모터, mU1a 프로모터, 신장 인자 1α 프로모터, 티록신 결합 글로불린 프로모터, 시냅신 프로모터, RNA 결합 Fox-1 동족체 3 프로모터, 칼슘/칼모듈린 의존성 단백질 키나아제 II 프로모터, 뉴런 특이적 에놀라아제 프로모터, 혈소판 유래 성장 인자 아단위 β, 소포성 글루타민산염 전달체 프로모터, 소마토스타틴 프로모터, 신경펩티드 Y 프로모터, 혈관작용 장관 펩티드 프로모터, 파브알부민 프로모터, 글루타민산염 탈카르복실화효소 65 프로모터, 글루타민산염 탈카르복실화효소 67 프로모터, 도파민 수용체 D1 프로모터, 도파민 수용체 D2 프로모터, 보체 C1q 유사 2 프로모터, 프로오피오멜라노코르틴 프로모터, 프로스페로 호메오복스 1 프로모터, 미소관 관련 단백질 1B 프로모터, 그리고 튜불린 알파 1 프로모터로 구성된 군에서 선택되는 프로모터를 포함하는, 발현 카세트.
- 청구항 제76항에 있어서, 발현 카세트는 서열 번호: 775, 777, 779, 781, 783-788, 796, 798-801, 803, 805, 807, 809, , 813, 817, 819 및 821 중에서 하나의 핵산 서열에 적어도 85% 서열 동일성을 갖는 폴리뉴클레오티드를 포함하는, 발현 카세트.
- 5'에서 3'로 다음을 포함하는 발현 카세트:
(a) 첫 번째 프로모터 서열;
(b) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나에 적어도 85% 서열 동일성을 갖는 첫 번째 가이드 뉴클레오티드 서열로서, 여기서 상기 첫 번째 가이드 뉴클레오티드 서열은 첫 번째 프로모터에 작동가능하게 연결되고;
(c) 두 번째 프로모터 서열;
(b) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나에 적어도 85% 서열 동일성을 갖는 두 번째 가이드 뉴클레오티드 서열로서, 여기서 상기 두 번째 가이드 뉴클레오티드 서열은 두 번째 프로모터에 작동가능하게 연결됨. - 청구항 제86항에 있어서, 첫 번째 가이드 뉴클레오티드 서열에 상보적이거나 또는 실질적으로 상보적인 첫 번째 승객 뉴클레오티드 서열을 더욱 포함하고, 여기서 첫 번째 승객 뉴클레오티드 서열은 첫 번째 가이드 뉴클레오티드 서열에 비하여 5' 또는 3'에 위치되는, 발현 카세트.
- 청구항 제86항 또는 제87항에 있어서, 두 번째 가이드 뉴클레오티드 서열에 상보적이거나 또는 실질적으로 상보적인 두 번째 승객 뉴클레오티드 서열을 더욱 포함하고, 여기서 두 번째 승객 뉴클레오티드 서열은 두 번째 가이드 뉴클레오티드 서열에 비하여 5' 또는 3'에 위치되는, 발현 카세트.
- 청구항 제86항 내지 제88항 중 어느 한 항에 있어서, 첫 번째 가이드 서열에 비하여 5' 에 위치된 첫 번째 5' 측부 영역을 더욱 포함하는, 발현 카세트.
- 청구항 제86항 내지 제89항 중 어느 한 항에 있어서, 첫 번째 가이드 서열에 비하여 3' 에 위치된 첫 번째 3' 측부 영역을 더욱 포함하는, 발현 카세트.
- 청구항 제86항 내지 제90항 중 어느 한 항에 있어서, 두 번째 가이드 서열에 비하여 5' 에 위치된 두 번째 5' 측부 영역을 더욱 포함하는, 발현 카세트.
- 청구항 제86항 내지 제91항 중 어느 한 항에 있어서, 두 번째 가이드 서열에 비하여 3' 에 위치된 두 번째 3' 측부 영역을 더욱 포함하는, 발현 카세트.
- 청구항 제86항 내지 제92항 중 어느 한 항에 있어서, 첫 번째 가이드 서열 및 첫 번째 승객 서열 사이에 위치된 첫 번째 루프 영역을 더욱 포함하고, 여기서 첫 번째 루프 영역은 첫 번째 마이크로RNA 루프 서열을 포함하는, 발현 카세트.
- 청구항 제86항 내지 제93항 중 어느 한 항에 있어서, 두 번째 가이드 서열 및 두 번째 승객 서열 사이에 위치된 두 번째 루프 영역을 더욱 포함하고, 여기서 두 번째 루프 영역은 두 번째 마이크로RNA 루프 서열을 포함하는, 발현 카세트.
- 청구항 제86항에 있어서, 발현 카세트는 서열 번호: 785-788 중에서 하나의 핵산 서열에 적어도 85% 서열 동일성을 갖는 폴리뉴클레오티드를 포함하는, 발현 카세트.
- 5'에서 3'로 다음을 포함하는 뉴클레오티드 서열을 포함하는 발현 카세트:
(a) 첫 번째 프로모터 서열;
(b) 첫 번째 승객 뉴클레오티드 서열의 5'에 위치된 첫 번째 5' 측부 영역;
(c) 첫 번째 스템 루프 서열로서, 5'에서 3'로:
(i) 첫 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 첫 번째 승객 뉴클레오티드 서열을 포함하는 첫 번째 5' 스템 루프 팔;
(ii) 첫 번째 루프 영역으로서, 여기서 상기 첫 번째 루프 영역은 첫 번째 마이크로RNA 루프 서열을 포함하고;
(iii) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나에 적어도 85% 서열 동일성을 갖는 첫 번째 가이드 뉴클레오티드 서열을 포함하는 첫 번째 3' 스템 루프 팔을 포함하는 첫 번째 스템 루프 서열;
(d) 상기 첫 번째 가이드 뉴클레오티드 서열의 3'에 위치된 첫 번째 3' 측부 영역;
(e) 두 번째 프로모터 서열;
(f) 두 번째 승객 뉴클레오티드 서열의 5'에 위치된 두 번째 5' 측부 영역;
(g) 두 번째 스템 루프 서열로서, 5'에서 3'로:
(i) 두 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 두 번째 승객 뉴클레오티드 서열을 포함하는 두 번째 5' 스템 루프 팔;
(ii) 두 번째 루프 영역으로서, 여기서 상기 두 번째 루프 영역은 두 번째 마이크로RNA 루프 서열을 포함하고;
(iii) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나에 적어도 85% 서열 동일성을 갖는 두 번째 가이드 뉴클레오티드 서열을 포함하는 두 번째 3' 스템 루프 팔을 포함하는 두 번째 스템 루프 서열;
그리고
(h) 상기 두 번째 가이드 뉴클레오티드 서열의 3'에 위치된 두 번째 3' 측부 영역. - 5'에서 3'로 다음을 포함하는 뉴클레오티드 서열을 포함하는 발현 카세트:
(a) 첫 번째 프로모터 서열;
(b) 첫 번째 승객 뉴클레오티드 서열의 5'에 위치된 첫 번째 5' 측부 영역;
(c) 첫 번째 스템 루프 서열로서, 5'에서 3'로:
(i) 첫 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 첫 번째 승객 뉴클레오티드 서열을 포함하는 첫 번째 5' 스템 루프 팔;
(ii) 첫 번째 루프 영역으로서, 여기서 상기 첫 번째 루프 영역은 첫 번째 마이크로RNA 루프 서열을 포함하고;
(iii) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나에 적어도 85% 서열 동일성을 갖는 첫 번째 가이드 뉴클레오티드 서열을 포함하는 첫 번째 3' 스템 루프 팔을 포함하는 첫 번째 스템 루프 서열;
(d) 상기 첫 번째 가이드 뉴클레오티드 서열의 3'에 위치된 첫 번째 3' 측부 영역;
(e) 두 번째 프로모터 서열;
(f) 두 번째 가이드 뉴클레오티드 서열의 5'에 위치된 두 번째 5' 측부 영역;
(g) 두 번째 스템 루프 서열로서, 5'에서 3'로:
(i) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나에 적어도 85% 서열 동일성을 갖는 가이드 뉴클레오티드 서열을 포함하는 두 번째 5' 스템 루프 팔;
(ii) 두 번째 루프 영역으로서, 여기서 상기 두 번째 루프 영역은 두 번째 마이크로RNA 루프 서열을 포함하고;
(iii) 두 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 두 번째 승객 뉴클레오티드 서열을 포함하는 두 번째 3' 스템 루프 팔을 포함하는 두 번째 스템 루프 서열;
그리고
(h) 상기 두 번째 승객 뉴클레오티드 서열의 3'에 위치된 두 번째 3' 측부 영역. - 5'에서 3'로 다음을 포함하는 뉴클레오티드 서열을 포함하는 발현 카세트:
(a) 첫 번째 프로모터 서열;
(b) 첫 번째 가이드 뉴클레오티드 서열의 5'에 위치된 첫 번째 5' 측부 영역;
(c) 첫 번째 스템 루프 서열로서, 5'에서 3'로:
(i) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나에 적어도 85% 서열 동일성을 갖는 첫 번째 가이드 뉴클레오티드 서열을 포함하는 첫 번째 5' 스템 루프 팔;
(ii) 첫 번째 루프 영역으로서, 여기서 상기 첫 번째 루프 영역은 첫 번째 마이크로RNA 루프 서열을 포함하고;
(iii) 첫 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 첫 번째 승객 뉴클레오티드 서열을 포함하는 첫 번째 3' 스템 루프 팔을 포함하는 첫 번째 스템 루프 서열;
(d) 상기 첫 번째 승객 뉴클레오티드 서열의 3'에 위치된 첫 번째 3' 측부 영역;
(e) 두 번째 프로모터 서열;
(f) 두 번째 승객 뉴클레오티드 서열의 5'에 위치된 두 번째 5' 측부 영역;
(g) 두 번째 스템 루프 서열로서, 5'에서 3'로:
(i) 두 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 두 번째 승객 뉴클레오티드 서열을 포함하는 두 번째 5' 스템 루프 팔;
(ii) 두 번째 루프 영역으로서, 여기서 상기 두 번째 루프 영역은 두 번째 마이크로RNA 루프 서열을 포함하고;
(iii) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나에 적어도 85% 서열 동일성을 갖는 두 번째 가이드 뉴클레오티드 서열을 포함하는 두 번째 3' 스템 루프 팔을 포함하는 두 번째 스템 루프 서열;
그리고
(h) 상기 두 번째 가이드 뉴클레오티드 서열의 3'에 위치된 두 번째 3' 측부 영역. - 5'에서 3'로 다음을 포함하는 뉴클레오티드 서열을 포함하는 발현 카세트:
(a) 첫 번째 프로모터 서열;
(b) 첫 번째 가이드 뉴클레오티드 서열의 5'에 위치된 첫 번째 5' 측부 영역;
(c) 첫 번째 스템 루프 서열로서, 5'에서 3'로:
(i) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나에 적어도 85% 서열 동일성을 갖는 첫 번째 가이드 뉴클레오티드 서열을 포함하는 첫 번째 5' 스템 루프 팔;
(ii) 첫 번째 루프 영역으로서, 여기서 상기 첫 번째 루프 영역은 첫 번째 마이크로RNA 루프 서열을 포함하고;
(iii) 첫 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 첫 번째 승객 뉴클레오티드 서열을 포함하는 첫 번째 3' 스템 루프 팔을 포함하는 첫 번째 스템 루프 서열;
(d) 상기 첫 번째 승객 뉴클레오티드 서열의 3'에 위치된 첫 번째 3' 측부 영역;
(e) 두 번째 프로모터 서열;
(f) 두 번째 가이드 뉴클레오티드 서열의 5'에 위치된 두 번째 5' 측부 영역;
(g) 두 번째 스템 루프 서열로서, 5'에서 3'로:
(i) 표 2 및/또는 표 3에서 열거된 가이드 서열 중에서 하나에 적어도 85% 서열 동일성을 갖는 가이드 뉴클레오티드 서열을 포함하는 두 번째 5' 스템 루프 팔;
(ii) 두 번째 루프 영역으로서, 여기서 상기 두 번째 루프 영역은 두 번째 마이크로RNA 루프 서열을 포함하고;
(iii) 두 번째 가이드 서열에 상보적이거나 또는 실질적으로 상보적인 두 번째 승객 뉴클레오티드 서열을 포함하는 두 번째 3' 스템 루프 팔을 포함하는 두 번째 스템 루프 서열;
그리고
(h) 상기 두 번째 승객 뉴클레오티드 서열의 3'에 위치된 두 번째 3' 측부 영역. - 청구항 제86항 내지 제99항 중 어느 한 항에 있어서, 첫 번째 프로모터 및/또는 두 번째 프로모터는 U6 프로모터, H1 프로모터, 7SK 프로모터, 아포지질단백질 E-인간 알파 1-항트립신 프로모터, CAG 프로모터, CBA 프로모터, CK8 프로모터, mU1a 프로모터, 신장 인자 1α 프로모터, 티록신 결합 글로불린 프로모터, 시냅신 프로모터, RNA 결합 Fox-1 동족체 3 프로모터, 칼슘/칼모듈린 의존성 단백질 키나아제 II 프로모터, 뉴런 특이적 에놀라아제 프로모터, 혈소판 유래 성장 인자 아단위 β, 소포성 글루타민산염 전달체 프로모터, 소마토스타틴 프로모터, 신경펩티드 Y 프로모터, 혈관작용 장관 펩티드 프로모터, 파브알부민 프로모터, 글루타민산염 탈카르복실화효소 65 프로모터, 글루타민산염 탈카르복실화효소 67 프로모터, 도파민 수용체 D1 프로모터, 도파민 수용체 D2 프로모터, 보체 C1q 유사 2 프로모터, 프로오피오멜라노코르틴 프로모터, 프로스페로 호메오복스 1 프로모터, 미소관 관련 단백질 1B 프로모터, 그리고 튜불린 알파 1 프로모터로 구성된 군에서 선택되는 프로모터에서 선택되는, 발현 카세트.
- 청구항 제86항 내지 제100항 중 어느 한 항에 있어서, 첫 번째 5' 측부 영역 및/또는 두 번째 5' 측부 영역은 서열 번호: 752, 754, 756, 759, 762, 765 또는 768 중에서 하나의 핵산 서열에 적어도 85% 서열 동일성을 갖는 폴리뉴클레오티드를 포함하는, 발현 카세트.
- 청구항 제86항 내지 제101항 중 어느 한 항에 있어서, 첫 번째 3' 측부 영역 및/또는 두 번째 3' 측부 영역은 서열 번호: 753, 755, 757, 760, 763, 766 또는 769 중에서 하나의 핵산 서열에 적어도 85% 서열 동일성을 갖는 폴리뉴클레오티드를 포함하는, 발현 카세트.
- 청구항 제86항 내지 제102항 중 어느 한 항에 있어서, 첫 번째 마이크로RNA 루프 서열 및/또는 두 번째 마이크로RNA 루프 서열은 miR-30, miR-155, miR-218-1, 또는 miR-124-3 서열인, 발현 카세트.
- 청구항 제103항에 있어서, 마이크로RNA 루프 서열은 서열 번호: 758, 761, 764, 767 또는 770 중에서 하나의 핵산 서열에 적어도 85% 서열 동일성을 갖는 폴리뉴클레오티드를 포함하는, 발현 카세트.
- 청구항 제74항 내지 제104항 중 어느 한 항에 있어서, 발현 카세트는 상기 발현 카세트의 5' 단부 상에서 5'-반전 말단 반복 (ITR) 서열 및 상기 발현 카세트의 3' 단부 상에서 3'-ITR 서열을 포함하는, 발현 카세트.
- 청구항 제105항에 있어서, 5'-ITR 및 3' ITR 서열은 AAV2 5'-ITR 및 3' ITR 서열인, 발현 카세트.
- 청구항 제105항 또는 제106항에 있어서, 5'-ITR 서열은 서열 번호: 746 또는 서열 번호: 747의 핵산 서열에 적어도 85% 서열 동일성을 갖는 폴리뉴클레오티드를 포함하는, 발현 카세트.
- 청구항 제105항 내지 제107항 중 어느 한 항에 있어서, 3'-ITR 서열은 서열 번호: 748, 서열 번호: 749, 또는 789의 핵산 서열에 적어도 85% 서열 동일성을 갖는 폴리뉴클레오티드를 포함하는, 발현 카세트.
- 청구항 제74항 내지 제108항 중 어느 한 항에 있어서, 인핸서 서열을 더욱 포함하는, 발현 카세트.
- 청구항 제109항에 있어서, 인핸서 서열은 서열 번호: 745의 핵산 서열에 적어도 85% 서열 동일성을 갖는 폴리뉴클레오티드를 포함하는, 발현 카세트.
- 청구항 제74항 내지 제110항 중 어느 한 항에 있어서, 인트론 서열을 더욱 포함하는, 발현 카세트.
- 청구항 제111항에 있어서, 인트론 서열은 서열 번호: 743 또는 서열 번호: 744의 핵산 서열에 적어도 85% 서열 동일성을 갖는 폴리뉴클레오티드를 포함하는, 발현 카세트.
- 청구항 제74항 내지 제112항 중 어느 한 항에 있어서, 하나 이상의 폴리아데닐화 신호를 더욱 포함하는, 발현 카세트.
- 청구항 제113항에 있어서, 하나 이상의 폴리아데닐화 신호는 토끼 베타-글로빈 (RBG) 폴리아데닐화 신호 또는 소 성장 호르몬 (BGH) 폴리아데닐화 신호인, 발현 카세트.
- 청구항 제114항에 있어서, RBG 폴리아데닐화 신호는 서열 번호: 750, 서열 번호: 751, 또는 서열 번호: 792의 핵산 서열에 적어도 85% 서열 동일성을 갖는 폴리뉴클레오티드를 포함하는, 발현 카세트.
- 청구항 제114항에 있어서, BGH 폴리아데닐화 신호는 서열 번호: 793의 핵산 서열에 적어도 85% 서열 동일성을 갖는 폴리뉴클레오티드를 포함하는, 발현 카세트.
- 청구항 제74항 내지 제116항 중 어느 한 항에 있어서, 발현 카세트는 청구항 제66항 내지 제73항 중 어느 한 항의 벡터 내로 통합되는, 발현 카세트.
- 청구항 제96항에 있어서, 발현 카세트는 서열 번호: 785, 서열 번호: 787, 또는 서열 번호: 788의 핵산 서열에 적어도 85% 서열 동일성을 갖는 폴리뉴클레오티드를 포함하는, 발현 카세트.
- 청구항 제98항에 있어서, 발현 카세트는 서열 번호: 786의 핵산 서열에 적어도 85% 서열 동일성을 갖는 폴리뉴클레오티드를 포함하는, 발현 카세트.
- 청구항 제74항 내지 제119항 중 어느 한 항에 있어서, 스터퍼 서열을 더욱 포함하는, 발현 카세트.
- 청구항 제120항에 있어서, 스터퍼 서열은 발현 카세트의 3' 단부에 배치되는, 발현 카세트.
- 청구항 제120항 또는 제121항에 있어서, 스터퍼 서열은 서열 번호: 815 또는 서열 번호: 816의 핵산 서열에 적어도 85% 서열 동일성을 갖고; 임의적으로 여기서 스터퍼 서열은 서열 번호: 815 또는 서열 번호: 816의 핵산 서열에 적어도 90% 서열 동일성을 갖고; 임의적으로 여기서 스터퍼 서열은 서열 번호: 815 또는 서열 번호: 816의 핵산 서열에 적어도 95% 서열 동일성을 갖고; 임의적으로 여기서 스터퍼 서열은 서열 번호: 815 또는 서열 번호: 816의 핵산 서열에 적어도 99% 서열 동일성을 갖고; 임의적으로 여기서 스터퍼 서열은 서열 번호: 815 또는 서열 번호: 816의 핵산 서열을 갖는, 발현 카세트.
- 세포에서 Grik2 발현을 저해하는 방법으로서, 상기 세포를 청구항 제1항 내지 제65항 중 어느 한 항의 적어도 하나의 상기 폴리뉴클레오티드, 청구항 제66항 내지 제73항 중 어느 한 항의 벡터, 또는 청구항 제74항 내지 제122항 중 어느 한 항의 발현 카세트와 접촉시키는 단계를 포함하는, 방법.
- 청구항 제123항에 있어서, 폴리뉴클레오티드는 Grik2 mRNA에 특이적으로 혼성화하고, 그리고 세포에서 Grik2 의 발현을 저해하거나 또는 감소시키는, 방법.
- 청구항 제123항 또는 제124항에 있어서, 상기 방법은 세포에서 GluK2 단백질의 수준을 감소시키는, 방법.
- 청구항 제125항에 있어서, 상기 방법은 세포에서 GluK2 단백질의 수준을 적어도 10%, 적어도 10%, 적어도 15%, 적어도 20%, 적어도 25%, 적어도 30%, 적어도 35%, 적어도 40%, 적어도 45%, 적어도 50%, 적어도 55%, 적어도 60%, 적어도 65%, 적어도 70%, 또는 적어도 75% 감소시키는, 방법.
- 청구항 제123항 내지 제126항 중 어느 한 항에 있어서, 세포는 뉴런인, 방법.
- 청구항 제127항에 있어서, 뉴런은 해마 뉴런인, 방법.
- 청구항 제128항에 있어서, 해마 뉴런은 DGC인, 방법.
- 청구항 제129항에 있어서, DGC는 일탈적 재발성 이끼 섬유 축삭돌기를 포함하는, 방법.
- 치료가 필요한 개체에서 장애를 치료하거나 또는 개선하는 방법으로서, 청구항 제1항 내지 제65항 중 어느 한 항의 적어도 하나의 상기 폴리뉴클레오티드, 청구항 제66항 내지 제73항 중 어느 한 항의 벡터, 또는 청구항 제74항 내지 제122항 중 어느 한 항의 발현 카세트를 상기 개체에게 투여하는 단계를 포함하는, 방법.
- 청구항 제131항에 있어서, 장애는 뇌전증인, 방법.
- 청구항 제132항에 있어서, 뇌전증은 측두엽 뇌전증 (TLE), 만성 뇌전증 및/또는 난치성 뇌전증인, 방법.
- 청구항 제133항에 있어서, 뇌전증은 TLE인, 방법.
- 청구항 제134항에 있어서, TLE는 편측 TLE (lTLE)인, 방법.
- 청구항 제134항에 있어서, TLE는 내측 TLE (mTLE)인, 방법.
- 청구항 제131항 내지 제136항 중 어느 한 항에 있어서, 개체는 인간인, 방법.
- 청구항 제1항 내지 제65항 중 어느 한 항의 폴리뉴클레오티드, 청구항 제66항 내지 제73항 중 어느 한 항의 벡터, 또는 청구항 제74항 내지 제122항 중 어느 한 항의 발현 카세트, 그리고 약학적으로 허용되는 운반체, 희석제 또는 부형제를 포함하는 약학적 조성물.
- 청구항 제138항의 약학적 조성물 및 포장 삽입물을 포함하는 키트.
- 청구항 제139항에 있어서, 포장 삽입물은 청구항 제131항 내지 제137항 중 어느 한 항의 방법에서 약학적 조성물의 사용법을 포함하는, 키트.
Applications Claiming Priority (7)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US202063050742P | 2020-07-10 | 2020-07-10 | |
| US63/050,742 | 2020-07-10 | ||
| US202163137669P | 2021-01-14 | 2021-01-14 | |
| US63/137,669 | 2021-01-14 | ||
| US202163185699P | 2021-05-07 | 2021-05-07 | |
| US63/185,699 | 2021-05-07 | ||
| PCT/US2021/041089 WO2022011262A1 (en) | 2020-07-10 | 2021-07-09 | Methods and compositions for treating epilepsy |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20230050336A true KR20230050336A (ko) | 2023-04-14 |
Family
ID=77168478
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020237004876A KR20230050336A (ko) | 2020-07-10 | 2021-07-09 | 뇌전증을 치료하기 위한 방법과 조성물 |
Country Status (10)
| Country | Link |
|---|---|
| US (1) | US20240018524A1 (ko) |
| EP (1) | EP4179091A1 (ko) |
| JP (1) | JP2023540429A (ko) |
| KR (1) | KR20230050336A (ko) |
| CN (1) | CN116113697A (ko) |
| AU (1) | AU2021305665A1 (ko) |
| BR (1) | BR112023000428A2 (ko) |
| CA (1) | CA3177613A1 (ko) |
| IL (1) | IL299771A (ko) |
| WO (1) | WO2022011262A1 (ko) |
Families Citing this family (4)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2022235614A2 (en) * | 2021-05-04 | 2022-11-10 | Regenxbio Inc. | Novel aav vectors and methods and uses thereof |
| US20230039652A1 (en) * | 2021-07-09 | 2023-02-09 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods for the treatment of epilepsy |
| WO2024054850A1 (en) * | 2022-09-06 | 2024-03-14 | The Trustees Of Princeton University | Rna-based compositions and methods of use thereof |
| WO2024079078A1 (en) * | 2022-10-10 | 2024-04-18 | Uniqure France | Methods and compositions for treating epilepsy |
Family Cites Families (162)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US414A (en) | 1837-09-28 | Moetise-latch foe | ||
| US941A (en) | 1838-09-22 | Machine for sawing shingles and staves | ||
| US5173A (en) | 1847-06-26 | Machinery for | ||
| US5139A (en) | 1847-06-05 | Cajsfkles | ||
| JPS5927900A (ja) | 1982-08-09 | 1984-02-14 | Wakunaga Seiyaku Kk | 固定化オリゴヌクレオチド |
| FR2540122B1 (fr) | 1983-01-27 | 1985-11-29 | Centre Nat Rech Scient | Nouveaux composes comportant une sequence d'oligonucleotide liee a un agent d'intercalation, leur procede de synthese et leur application |
| US4605735A (en) | 1983-02-14 | 1986-08-12 | Wakunaga Seiyaku Kabushiki Kaisha | Oligonucleotide derivatives |
| US4948882A (en) | 1983-02-22 | 1990-08-14 | Syngene, Inc. | Single-stranded labelled oligonucleotides, reactive monomers and methods of synthesis |
| US4824941A (en) | 1983-03-10 | 1989-04-25 | Julian Gordon | Specific antibody to the native form of 2'5'-oligonucleotides, the method of preparation and the use as reagents in immunoassays or for binding 2'5'-oligonucleotides in biological systems |
| US4587044A (en) | 1983-09-01 | 1986-05-06 | The Johns Hopkins University | Linkage of proteins to nucleic acids |
| US5118802A (en) | 1983-12-20 | 1992-06-02 | California Institute Of Technology | DNA-reporter conjugates linked via the 2' or 5'-primary amino group of the 5'-terminal nucleoside |
| US5430136A (en) | 1984-10-16 | 1995-07-04 | Chiron Corporation | Oligonucleotides having selectably cleavable and/or abasic sites |
| US5258506A (en) | 1984-10-16 | 1993-11-02 | Chiron Corporation | Photolabile reagents for incorporation into oligonucleotide chains |
| US4828979A (en) | 1984-11-08 | 1989-05-09 | Life Technologies, Inc. | Nucleotide analogs for nucleic acid labeling and detection |
| US4897355A (en) | 1985-01-07 | 1990-01-30 | Syntex (U.S.A.) Inc. | N[ω,(ω-1)-dialkyloxy]- and N-[ω,(ω-1)-dialkenyloxy]-alk-1-yl-N,N,N-tetrasubstituted ammonium lipids and uses therefor |
| US4683202A (en) | 1985-03-28 | 1987-07-28 | Cetus Corporation | Process for amplifying nucleic acid sequences |
| US4762779A (en) | 1985-06-13 | 1988-08-09 | Amgen Inc. | Compositions and methods for functionalizing nucleic acids |
| US5139941A (en) | 1985-10-31 | 1992-08-18 | University Of Florida Research Foundation, Inc. | AAV transduction vectors |
| US5317098A (en) | 1986-03-17 | 1994-05-31 | Hiroaki Shizuya | Non-radioisotope tagging of fragments |
| JPS638396A (ja) | 1986-06-30 | 1988-01-14 | Wakunaga Pharmaceut Co Ltd | ポリ標識化オリゴヌクレオチド誘導体 |
| US4837028A (en) | 1986-12-24 | 1989-06-06 | Liposome Technology, Inc. | Liposomes with enhanced circulation time |
| US4920016A (en) | 1986-12-24 | 1990-04-24 | Linear Technology, Inc. | Liposomes with enhanced circulation time |
| US4904582A (en) | 1987-06-11 | 1990-02-27 | Synthetic Genetics | Novel amphiphilic nucleic acid conjugates |
| US5585481A (en) | 1987-09-21 | 1996-12-17 | Gen-Probe Incorporated | Linking reagents for nucleotide probes |
| US5525465A (en) | 1987-10-28 | 1996-06-11 | Howard Florey Institute Of Experimental Physiology And Medicine | Oligonucleotide-polyamide conjugates and methods of production and applications of the same |
| DE3738460A1 (de) | 1987-11-12 | 1989-05-24 | Max Planck Gesellschaft | Modifizierte oligonukleotide |
| US5082830A (en) | 1988-02-26 | 1992-01-21 | Enzo Biochem, Inc. | End labeled nucleotide probe |
| US5109124A (en) | 1988-06-01 | 1992-04-28 | Biogen, Inc. | Nucleic acid probe linked to a label having a terminal cysteine |
| US5262536A (en) | 1988-09-15 | 1993-11-16 | E. I. Du Pont De Nemours And Company | Reagents for the preparation of 5'-tagged oligonucleotides |
| US5512439A (en) | 1988-11-21 | 1996-04-30 | Dynal As | Oligonucleotide-linked magnetic particles and uses thereof |
| US5457183A (en) | 1989-03-06 | 1995-10-10 | Board Of Regents, The University Of Texas System | Hydroxylated texaphyrins |
| US5599923A (en) | 1989-03-06 | 1997-02-04 | Board Of Regents, University Of Tx | Texaphyrin metal complexes having improved functionalization |
| FR2645866B1 (fr) | 1989-04-17 | 1991-07-05 | Centre Nat Rech Scient | Nouvelles lipopolyamines, leur preparation et leur emploi |
| US5391723A (en) | 1989-05-31 | 1995-02-21 | Neorx Corporation | Oligonucleotide conjugates |
| US4958013A (en) | 1989-06-06 | 1990-09-18 | Northwestern University | Cholesteryl modified oligonucleotides |
| US5143854A (en) | 1989-06-07 | 1992-09-01 | Affymax Technologies N.V. | Large scale photolithographic solid phase synthesis of polypeptides and receptor binding screening thereof |
| US5744101A (en) | 1989-06-07 | 1998-04-28 | Affymax Technologies N.V. | Photolabile nucleoside protecting groups |
| US5451463A (en) | 1989-08-28 | 1995-09-19 | Clontech Laboratories, Inc. | Non-nucleoside 1,3-diol reagents for labeling synthetic oligonucleotides |
| US5254469A (en) | 1989-09-12 | 1993-10-19 | Eastman Kodak Company | Oligonucleotide-enzyme conjugate that can be used as a probe in hybridization assays and polymerase chain reaction procedures |
| US5292873A (en) | 1989-11-29 | 1994-03-08 | The Research Foundation Of State University Of New York | Nucleic acids labeled with naphthoquinone probe |
| US5486603A (en) | 1990-01-08 | 1996-01-23 | Gilead Sciences, Inc. | Oligonucleotide having enhanced binding affinity |
| US7037646B1 (en) | 1990-01-11 | 2006-05-02 | Isis Pharmaceuticals, Inc. | Amine-derivatized nucleosides and oligonucleosides |
| US5578718A (en) | 1990-01-11 | 1996-11-26 | Isis Pharmaceuticals, Inc. | Thiol-derivatized nucleosides |
| US6783931B1 (en) | 1990-01-11 | 2004-08-31 | Isis Pharmaceuticals, Inc. | Amine-derivatized nucleosides and oligonucleosides |
| AU7579991A (en) | 1990-02-20 | 1991-09-18 | Gilead Sciences, Inc. | Pseudonucleosides and pseudonucleotides and their polymers |
| US5214136A (en) | 1990-02-20 | 1993-05-25 | Gilead Sciences, Inc. | Anthraquinone-derivatives oligonucleotides |
| US5264618A (en) | 1990-04-19 | 1993-11-23 | Vical, Inc. | Cationic lipids for intracellular delivery of biologically active molecules |
| DE69032425T2 (de) | 1990-05-11 | 1998-11-26 | Microprobe Corp., Bothell, Wash. | Teststreifen zum Eintauchen für Nukleinsäure-Hybridisierungsassays und Verfahren zur kovalenten Immobilisierung von Oligonucleotiden |
| AU8200191A (en) | 1990-07-09 | 1992-02-04 | United States of America, as represented by the Secretary, U.S. Department of Commerce, The | High efficiency packaging of mutant adeno-associated virus using amber suppressions |
| US5138045A (en) | 1990-07-27 | 1992-08-11 | Isis Pharmaceuticals | Polyamine conjugated oligonucleotides |
| US5218105A (en) | 1990-07-27 | 1993-06-08 | Isis Pharmaceuticals | Polyamine conjugated oligonucleotides |
| US5608046A (en) | 1990-07-27 | 1997-03-04 | Isis Pharmaceuticals, Inc. | Conjugated 4'-desmethyl nucleoside analog compounds |
| US5688941A (en) | 1990-07-27 | 1997-11-18 | Isis Pharmaceuticals, Inc. | Methods of making conjugated 4' desmethyl nucleoside analog compounds |
| US5245022A (en) | 1990-08-03 | 1993-09-14 | Sterling Drug, Inc. | Exonuclease resistant terminally substituted oligonucleotides |
| US5512667A (en) | 1990-08-28 | 1996-04-30 | Reed; Michael W. | Trifunctional intermediates for preparing 3'-tailed oligonucleotides |
| US5173414A (en) | 1990-10-30 | 1992-12-22 | Applied Immune Sciences, Inc. | Production of recombinant adeno-associated virus vectors |
| EP0556301B1 (en) | 1990-11-08 | 2001-01-10 | Hybridon, Inc. | Incorporation of multiple reporter groups on synthetic oligonucleotides |
| US5766855A (en) | 1991-05-24 | 1998-06-16 | Buchardt, Deceased; Ole | Peptide nucleic acids having enhanced binding affinity and sequence specificity |
| US5539082A (en) | 1993-04-26 | 1996-07-23 | Nielsen; Peter E. | Peptide nucleic acids |
| DK51092D0 (da) | 1991-05-24 | 1992-04-15 | Ole Buchardt | Oligonucleotid-analoge betegnet pna, monomere synthoner og fremgangsmaade til fremstilling deraf samt anvendelser deraf |
| US5371241A (en) | 1991-07-19 | 1994-12-06 | Pharmacia P-L Biochemicals Inc. | Fluorescein labelled phosphoramidites |
| JP3534749B2 (ja) | 1991-08-20 | 2004-06-07 | アメリカ合衆国 | アデノウイルスが介在する胃腸管への遺伝子の輸送 |
| US5283185A (en) | 1991-08-28 | 1994-02-01 | University Of Tennessee Research Corporation | Method for delivering nucleic acids into cells |
| US5677195A (en) | 1991-11-22 | 1997-10-14 | Affymax Technologies N.V. | Combinatorial strategies for polymer synthesis |
| US5595726A (en) | 1992-01-21 | 1997-01-21 | Pharmacyclics, Inc. | Chromophore probe for detection of nucleic acid |
| US5565552A (en) | 1992-01-21 | 1996-10-15 | Pharmacyclics, Inc. | Method of expanded porphyrin-oligonucleotide conjugate synthesis |
| CA2134773A1 (en) | 1992-06-04 | 1993-12-09 | Robert J. Debs | Methods and compositions for in vivo gene therapy |
| AU4541093A (en) | 1992-06-18 | 1994-01-24 | Genpharm International, Inc. | Methods for producing transgenic non-human animals harboring a yeast artificial chromosome |
| US5272250A (en) | 1992-07-10 | 1993-12-21 | Spielvogel Bernard F | Boronated phosphoramidate compounds |
| DE69329503T2 (de) | 1992-11-13 | 2001-05-03 | Idec Pharma Corp | Therapeutische Verwendung von chimerischen und markierten Antikörpern, die gegen ein Differenzierung-Antigen gerichtet sind, dessen Expression auf menschliche B Lymphozyt beschränkt ist, für die Behandlung von B-Zell-Lymphoma |
| US5869305A (en) | 1992-12-04 | 1999-02-09 | The University Of Pittsburgh | Recombinant viral vector system |
| US5574142A (en) | 1992-12-15 | 1996-11-12 | Microprobe Corporation | Peptide linkers for improved oligonucleotide delivery |
| US6294664B1 (en) | 1993-07-29 | 2001-09-25 | Isis Pharmaceuticals, Inc. | Synthesis of oligonucleotides |
| US5539083A (en) | 1994-02-23 | 1996-07-23 | Isis Pharmaceuticals, Inc. | Peptide nucleic acid combinatorial libraries and improved methods of synthesis |
| US5543152A (en) | 1994-06-20 | 1996-08-06 | Inex Pharmaceuticals Corporation | Sphingosomes for enhanced drug delivery |
| US6204059B1 (en) | 1994-06-30 | 2001-03-20 | University Of Pittsburgh | AAV capsid vehicles for molecular transfer |
| US5597696A (en) | 1994-07-18 | 1997-01-28 | Becton Dickinson And Company | Covalent cyanine dye oligonucleotide conjugates |
| US5580731A (en) | 1994-08-25 | 1996-12-03 | Chiron Corporation | N-4 modified pyrimidine deoxynucleotides and oligonucleotide probes synthesized therewith |
| US5556752A (en) | 1994-10-24 | 1996-09-17 | Affymetrix, Inc. | Surface-bound, unimolecular, double-stranded DNA |
| CA2220950A1 (en) | 1995-05-26 | 1996-11-28 | Somatix Therapy Corporation | Delivery vehicles comprising stable lipid/nucleic acid complexes |
| US7422902B1 (en) | 1995-06-07 | 2008-09-09 | The University Of British Columbia | Lipid-nucleic acid particles prepared via a hydrophobic lipid-nucleic acid complex intermediate and use for gene transfer |
| CA2222328C (en) | 1995-06-07 | 2012-01-10 | Inex Pharmaceuticals Corporation | Lipid-nucleic acid particles prepared via a hydrophobic lipid-nucleic acid complex intermediate and use for gene transfer |
| US5545531A (en) | 1995-06-07 | 1996-08-13 | Affymax Technologies N.V. | Methods for making a device for concurrently processing multiple biological chip assays |
| US5981501A (en) | 1995-06-07 | 1999-11-09 | Inex Pharmaceuticals Corp. | Methods for encapsulating plasmids in lipid bilayers |
| US6001650A (en) | 1995-08-03 | 1999-12-14 | Avigen, Inc. | High-efficiency wild-type-free AAV helper functions |
| US5801030A (en) | 1995-09-01 | 1998-09-01 | Genvec, Inc. | Methods and vectors for site-specific recombination |
| US6013516A (en) | 1995-10-06 | 2000-01-11 | The Salk Institute For Biological Studies | Vector and method of use for nucleic acid delivery to non-dividing cells |
| US5858397A (en) | 1995-10-11 | 1999-01-12 | University Of British Columbia | Liposomal formulations of mitoxantrone |
| US5854033A (en) | 1995-11-21 | 1998-12-29 | Yale University | Rolling circle replication reporter systems |
| US6043060A (en) | 1996-11-18 | 2000-03-28 | Imanishi; Takeshi | Nucleotide analogues |
| US6576752B1 (en) | 1997-02-14 | 2003-06-10 | Isis Pharmaceuticals, Inc. | Aminooxy functionalized oligomers |
| US6034135A (en) | 1997-03-06 | 2000-03-07 | Promega Biosciences, Inc. | Dimeric cationic lipids |
| US6770748B2 (en) | 1997-03-07 | 2004-08-03 | Takeshi Imanishi | Bicyclonucleoside and oligonucleotide analogue |
| JP3756313B2 (ja) | 1997-03-07 | 2006-03-15 | 武 今西 | 新規ビシクロヌクレオシド及びオリゴヌクレオチド類縁体 |
| DE04020014T1 (de) | 1997-09-12 | 2006-01-26 | Exiqon A/S | Bi-zyklische - Nukleosid,Nnukleotid und Oligonukleotid-Analoga |
| US6136597A (en) | 1997-09-18 | 2000-10-24 | The Salk Institute For Biological Studies | RNA export element |
| US6639051B2 (en) | 1997-10-20 | 2003-10-28 | Curis, Inc. | Regulation of epithelial tissue by hedgehog-like polypeptides, and formulations and uses related thereto |
| US5994136A (en) | 1997-12-12 | 1999-11-30 | Cell Genesys, Inc. | Method and means for producing high titer, safe, recombinant lentivirus vectors |
| US6506559B1 (en) | 1997-12-23 | 2003-01-14 | Carnegie Institute Of Washington | Genetic inhibition by double-stranded RNA |
| US6320017B1 (en) | 1997-12-23 | 2001-11-20 | Inex Pharmaceuticals Corp. | Polyamide oligomers |
| US6649881B2 (en) | 1998-06-04 | 2003-11-18 | American Water Heater Company | Electric water heater with pulsed electronic control and detection |
| US6566131B1 (en) | 2000-10-04 | 2003-05-20 | Isis Pharmaceuticals, Inc. | Antisense modulation of Smad6 expression |
| EP1096921B1 (en) | 1998-07-20 | 2003-04-16 | Protiva Biotherapeutics Inc. | Liposomal encapsulated nucleic acid-complexes |
| US6410323B1 (en) | 1999-08-31 | 2002-06-25 | Isis Pharmaceuticals, Inc. | Antisense modulation of human Rho family gene expression |
| US6107091A (en) | 1998-12-03 | 2000-08-22 | Isis Pharmaceuticals Inc. | Antisense inhibition of G-alpha-16 expression |
| US5981732A (en) | 1998-12-04 | 1999-11-09 | Isis Pharmaceuticals Inc. | Antisense modulation of G-alpha-13 expression |
| TR200604211T1 (tr) | 1999-02-12 | 2007-02-21 | Daiichi Sankyo Company Limiteddaiichi Sankyo Company Limited | Yeni nükleosid ve oligonükleotid analoglarıYeni nükleosid ve oligonükleotid analogları |
| WO2000056748A1 (en) | 1999-03-18 | 2000-09-28 | Exiqon A/S | Xylo-lna analogues |
| ES2269113T3 (es) | 1999-03-24 | 2007-04-01 | Exiqon A/S | Sintesis mejorada de -2.2.1 / biciclo-nucleosidos. |
| US6046321A (en) | 1999-04-09 | 2000-04-04 | Isis Pharmaceuticals Inc. | Antisense modulation of G-alpha-i1 expression |
| CA2372085C (en) | 1999-05-04 | 2009-10-27 | Exiqon A/S | L-ribo-lna analogues |
| DE60039766D1 (de) | 1999-08-09 | 2008-09-18 | Targeted Genetics Corp | Terologen nukleotidsequenz von einem rekombinanten viralen vektor durch ausgestaltung der sequenz in einer art und weise, dass basenpaarungen innerhalb der sequenz entstehen |
| IL148916A0 (en) | 1999-10-04 | 2002-09-12 | Exiqon As | Design of high affinity rnase h recruiting oligonucleotide |
| GB9927444D0 (en) | 1999-11-19 | 2000-01-19 | Cancer Res Campaign Tech | Inhibiting gene expression |
| AU2001245793A1 (en) | 2000-03-16 | 2001-09-24 | Cold Spring Harbor Laboratory | Methods and compositions for rna interference |
| US6365354B1 (en) | 2000-07-31 | 2002-04-02 | Isis Pharmaceuticals, Inc. | Antisense modulation of lysophospholipase I expression |
| EP1334109B1 (en) | 2000-10-04 | 2006-05-10 | Santaris Pharma A/S | Improved synthesis of purine locked nucleic acid analogues |
| US6566135B1 (en) | 2000-10-04 | 2003-05-20 | Isis Pharmaceuticals, Inc. | Antisense modulation of caspase 6 expression |
| AU2002317437A1 (en) | 2001-05-18 | 2002-12-03 | Cureon A/S | Therapeutic uses of lna-modified oligonucleotides in infectious diseases |
| DE60202681T2 (de) | 2001-07-12 | 2006-01-12 | Santaris Pharma A/S | Verfahren zur herstellung des lna phosphoramidite |
| NZ618298A (en) | 2001-11-13 | 2015-04-24 | Univ Pennsylvania | A method of detecting and/or identifying adeno-associated virus (aav) sequences and isolating novel sequences identified thereby |
| ES2975413T3 (es) | 2001-12-17 | 2024-07-05 | Univ Pennsylvania | Secuencias de serotipo 8 de virus adenoasociado (AAV), vectores que las contienen y usos de las mismas |
| JP4269260B2 (ja) | 2003-06-05 | 2009-05-27 | 三浦工業株式会社 | バルブ |
| JP5054975B2 (ja) | 2003-09-30 | 2012-10-24 | ザ・トラステイーズ・オブ・ザ・ユニバーシテイ・オブ・ペンシルベニア | アデノ随伴ウイルス(aav)の同源系統群、配列、それらを含有するベクターおよびそれらの用途 |
| DK1706489T3 (da) | 2003-12-23 | 2010-09-13 | Santaris Pharma As | Oligomer forbindelser for modulationen af BCL-2 |
| JP2007527240A (ja) | 2004-03-01 | 2007-09-27 | マサチューセッツ インスティテュート オブ テクノロジー | アレルギー性鼻炎および喘息のためのRNAiベースの治療 |
| WO2005116204A1 (ja) * | 2004-05-11 | 2005-12-08 | Rnai Co., Ltd. | Rna干渉を生じさせるポリヌクレオチド、および、これを用いた遺伝子発現抑制方法 |
| US20070004624A1 (en) * | 2004-06-16 | 2007-01-04 | Luc Van Rompaey | Methods for modulating bone tissue formation, orthogenic agents and pharmaceutical compositions |
| US7183969B2 (en) | 2004-12-22 | 2007-02-27 | Raytheon Company | System and technique for calibrating radar arrays |
| CN117363655A (zh) | 2005-04-07 | 2024-01-09 | 宾夕法尼亚大学托管会 | 增强腺相关病毒载体功能的方法 |
| US7456683B2 (en) | 2005-06-09 | 2008-11-25 | Panasonic Corporation | Amplitude error compensating device and quadrature skew error compensating device |
| CA2708173C (en) | 2007-12-04 | 2016-02-02 | Alnylam Pharmaceuticals, Inc. | Targeting lipids |
| ES2929031T3 (es) | 2008-02-19 | 2022-11-24 | Uniqure Ip Bv | Optimización de la expresión de las proteínas rep y cap parvovirales en células de insecto |
| JP6174318B2 (ja) | 2009-04-30 | 2017-08-02 | ザ・トラステイーズ・オブ・ザ・ユニバーシテイ・オブ・ペンシルベニア | アデノ随伴ウイルス構築物を含んでなるコンダクティング気道細胞を標的とするための組成物 |
| US8734809B2 (en) | 2009-05-28 | 2014-05-27 | University Of Massachusetts | AAV's and uses thereof |
| PL2440183T3 (pl) | 2009-06-10 | 2019-01-31 | Arbutus Biopharma Corporation | Ulepszona formulacja lipidowa |
| US8628966B2 (en) | 2010-04-30 | 2014-01-14 | City Of Hope | CD34-derived recombinant adeno-associated vectors for stem cell transduction and systemic therapeutic gene transfer |
| US8927514B2 (en) | 2010-04-30 | 2015-01-06 | City Of Hope | Recombinant adeno-associated vectors for targeted treatment |
| CN103189507A (zh) | 2010-10-27 | 2013-07-03 | 学校法人自治医科大学 | 用于向神经系统细胞导入基因的腺相关病毒粒子 |
| US9409953B2 (en) | 2011-02-10 | 2016-08-09 | The University Of North Carolina At Chapel Hill | Viral vectors with modified transduction profiles and methods of making and using the same |
| PL2699270T3 (pl) | 2011-04-22 | 2017-12-29 | The Regents Of The University Of California | Wiriony wirusa towarzyszącego adenowirusom z różnymi kapsydami i sposoby ich zastosowania |
| EP3564393A1 (en) | 2011-06-21 | 2019-11-06 | Alnylam Pharmaceuticals, Inc. | Assays and methods for determining activity of a therapeutic agent in a subject |
| WO2013029030A1 (en) | 2011-08-24 | 2013-02-28 | The Board Of Trustees Of The Leland Stanford Junior University | New aav capsid proteins for nucleic acid transfer |
| EP3470523A1 (en) | 2012-05-09 | 2019-04-17 | Oregon Health & Science University | Adeno associated virus plasmids and vectors |
| CA2905952A1 (en) | 2013-03-13 | 2014-10-02 | The Children's Hospital Of Philadelphia | Adeno-associated virus vectors and methods of use thereof |
| MX2015014712A (es) | 2013-04-20 | 2016-04-27 | Res Inst Nationwide Childrens Hospital | Administración de virus adeno-asociado recombinante de construcciones de polinucléotidos u7snrna dirigida al exon 2. |
| PL2992098T3 (pl) | 2013-05-01 | 2019-09-30 | Ionis Pharmaceuticals, Inc. | Kompozycje i sposoby modulowania ekspresji hbv i ttr |
| WO2014195754A1 (en) | 2013-06-05 | 2014-12-11 | Institut National De La Sante Et De La Recherche Medicale (Inserm) | Hydrophobically modified antisense oligonucleotides comprising a triple alkyl chain |
| WO2014195755A1 (en) | 2013-06-05 | 2014-12-11 | Institut National De La Sante Et De La Recherche Medicale (Inserm) | Hydrophobically modified antisense oligonucleotides comprising a ketal group |
| KR102380265B1 (ko) | 2013-07-22 | 2022-03-29 | 더 칠드런스 호스피탈 오브 필라델피아 | 변종 aav 및 조성물, 세포, 기관 및 조직으로의 유전자 전이를 위한 방법 및 용도 |
| WO2015038958A1 (en) | 2013-09-13 | 2015-03-19 | California Institute Of Technology | Selective recovery |
| US10016424B2 (en) * | 2013-09-16 | 2018-07-10 | INSERM (Institut National de la Santé et de la Recherche Médicale | Method and pharmaceutical composition for use in the treatment of epilepsy |
| CN115141258A (zh) | 2013-10-11 | 2022-10-04 | 马萨诸塞眼科耳科诊所 | 预测祖先病毒序列的方法及其用途 |
| WO2015073360A2 (en) | 2013-11-12 | 2015-05-21 | New England Biolabs Inc. | Dnmt inhibitors |
| US10746742B2 (en) | 2014-04-25 | 2020-08-18 | Oregon Health & Science University | Methods of viral neutralizing antibody epitope mapping |
| WO2015191508A1 (en) | 2014-06-09 | 2015-12-17 | Voyager Therapeutics, Inc. | Chimeric capsids |
| SG10201902574RA (en) | 2014-09-24 | 2019-04-29 | Hope City | Adeno-associated virus vector variants for high efficiency genome editing and methods thereof |
| JP6627515B2 (ja) | 2015-02-06 | 2020-01-08 | Jnc株式会社 | 3,6−ジヒドロ−2h−ピランを有する誘電率異方性が負の液晶性化合物、液晶組成物および液晶表示素子 |
| JP6665466B2 (ja) | 2015-09-26 | 2020-03-13 | 日亜化学工業株式会社 | 半導体発光素子及びその製造方法 |
| WO2017070491A1 (en) | 2015-10-23 | 2017-04-27 | Applied Genetic Technologies Corporation | Ophthalmic formulations |
| BR112019021852A2 (pt) | 2017-04-18 | 2020-06-02 | Alnylam Pharmaceuticals, Inc. | Agente de rnai e uma vacina contra hbv, uso ou método e kit para tratamento |
| WO2021005223A1 (en) * | 2019-07-10 | 2021-01-14 | INSERM (Institut National de la Santé et de la Recherche Médicale) | Methods for the treatment of epilepsy |
-
2021
- 2021-07-09 CA CA3177613A patent/CA3177613A1/en active Pending
- 2021-07-09 JP JP2023501422A patent/JP2023540429A/ja active Pending
- 2021-07-09 EP EP21749482.2A patent/EP4179091A1/en active Pending
- 2021-07-09 KR KR1020237004876A patent/KR20230050336A/ko active Search and Examination
- 2021-07-09 IL IL299771A patent/IL299771A/en unknown
- 2021-07-09 AU AU2021305665A patent/AU2021305665A1/en active Pending
- 2021-07-09 WO PCT/US2021/041089 patent/WO2022011262A1/en active Application Filing
- 2021-07-09 US US18/014,906 patent/US20240018524A1/en active Pending
- 2021-07-09 BR BR112023000428A patent/BR112023000428A2/pt unknown
- 2021-07-09 CN CN202180054648.5A patent/CN116113697A/zh active Pending
Also Published As
| Publication number | Publication date |
|---|---|
| US20240018524A1 (en) | 2024-01-18 |
| CN116113697A (zh) | 2023-05-12 |
| CA3177613A1 (en) | 2022-04-13 |
| AU2021305665A1 (en) | 2023-02-23 |
| BR112023000428A2 (pt) | 2023-03-14 |
| EP4179091A1 (en) | 2023-05-17 |
| IL299771A (en) | 2023-03-01 |
| WO2022011262A1 (en) | 2022-01-13 |
| JP2023540429A (ja) | 2023-09-25 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| KR102306797B1 (ko) | Pd-l1 발현의 감소를 위한 올리고뉴클레오티드 | |
| KR102366078B1 (ko) | Pkk 발현을 조절하는 조성물 및 방법 | |
| KR102356388B1 (ko) | 안지오포이에틴-유사 3 발현을 조절하기 위한 조성물 및 방법 | |
| KR20230050336A (ko) | 뇌전증을 치료하기 위한 방법과 조성물 | |
| JP7416451B2 (ja) | CRISPR-Casによる標的化された核内RNA切断及びポリアデニル化 | |
| EP1799826B1 (en) | siRNA-MEDIATED GENE SILENCING OF ALPHA SYNUCLEIN | |
| US20240294911A1 (en) | INSULIN-LIKE GROWTH FACTOR BINDING PROTEIN, ACID LABILE SUBUNIT (IGFALS) AND INSULIN-LIKE GROWTH FACTOR 1 (IGF-1) iRNA COMPOSITIONS AND METHODS OF USE THEREOF | |
| KR20150006477A (ko) | 단일-뉴클레오타이드 kras 돌연변이에 특이적인 이-기능성 짧은-헤어핀 rna (bi-shrna) | |
| KR20200058509A (ko) | Attr 아밀로이드증의 ttr 유전자 편집 및 치료를 위한 조성물 및 방법 | |
| CN114174520A (zh) | 用于选择性基因调节的组合物和方法 | |
| KR20210099090A (ko) | Msh3 활성과 연관된 트리뉴클레오티드 반복 확장 장애의 치료 방법 | |
| KR20230079405A (ko) | Snca-관련 신경퇴행성 질환의 치료 또는 예방을 위한 snca irna 조성물 및 그 사용 방법 | |
| US20230227829A1 (en) | Methods and compositions for treating epilepsy | |
| KR20230003478A (ko) | 비-바이러스성 dna 벡터 및 고셰 치료제 발현을 위한 이의 용도 | |
| KR20240042363A (ko) | 뇌전증을 치료하기 위한 방법 및 조성물 | |
| KR20240099164A (ko) | Pah-조절 조성물 및 방법 | |
| CN117203336A (zh) | 亨廷顿蛋白(HTT)iRNA药剂组合物以及其使用方法 | |
| CN117677704A (zh) | 用于治疗癫痫的方法和组合物 | |
| WO2023137260A2 (en) | Bancr compositions and methods for treating disease | |
| KR20240010467A (ko) | 인간 미토콘드리아 게놈을 표적화하는 조작된 메가뉴클레아제 | |
| CN117795071A (zh) | 用于通过肝配蛋白-b2抑制防止由内皮功能障碍导致的肾功能障碍的反义寡核苷酸 | |
| WO2023019185A1 (en) | Compositions and methods for engineering stable tregs |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A201 | Request for examination |