KR20160035077A - 플라스미노겐 활성인자 저해제-1(pai-1)에 대한 항체 및 그의 용도 - Google Patents
플라스미노겐 활성인자 저해제-1(pai-1)에 대한 항체 및 그의 용도 Download PDFInfo
- Publication number
- KR20160035077A KR20160035077A KR1020167005652A KR20167005652A KR20160035077A KR 20160035077 A KR20160035077 A KR 20160035077A KR 1020167005652 A KR1020167005652 A KR 1020167005652A KR 20167005652 A KR20167005652 A KR 20167005652A KR 20160035077 A KR20160035077 A KR 20160035077A
- Authority
- KR
- South Korea
- Prior art keywords
- seq
- region
- heavy chain
- light chain
- pai
- Prior art date
Links
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/38—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against protease inhibitors of peptide structure
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/395—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P1/00—Drugs for disorders of the alimentary tract or the digestive system
- A61P1/16—Drugs for disorders of the alimentary tract or the digestive system for liver or gallbladder disorders, e.g. hepatoprotective agents, cholagogues, litholytics
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P11/00—Drugs for disorders of the respiratory system
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P13/00—Drugs for disorders of the urinary system
- A61P13/12—Drugs for disorders of the urinary system of the kidneys
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P17/00—Drugs for dermatological disorders
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P7/00—Drugs for disorders of the blood or the extracellular fluid
- A61P7/02—Antithrombotic agents; Anticoagulants; Platelet aggregation inhibitors
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P9/00—Drugs for disorders of the cardiovascular system
- A61P9/10—Drugs for disorders of the cardiovascular system for treating ischaemic or atherosclerotic diseases, e.g. antianginal drugs, coronary vasodilators, drugs for myocardial infarction, retinopathy, cerebrovascula insufficiency, renal arteriosclerosis
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/505—Medicinal preparations containing antigens or antibodies comprising antibodies
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/20—Immunoglobulins specific features characterized by taxonomic origin
- C07K2317/24—Immunoglobulins specific features characterized by taxonomic origin containing regions, domains or residues from different species, e.g. chimeric, humanized or veneered
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/30—Immunoglobulins specific features characterized by aspects of specificity or valency
- C07K2317/32—Immunoglobulins specific features characterized by aspects of specificity or valency specific for a neo-epitope on a complex, e.g. antibody-antigen or ligand-receptor
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/30—Immunoglobulins specific features characterized by aspects of specificity or valency
- C07K2317/34—Identification of a linear epitope shorter than 20 amino acid residues or of a conformational epitope defined by amino acid residues
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/56—Immunoglobulins specific features characterized by immunoglobulin fragments variable (Fv) region, i.e. VH and/or VL
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/56—Immunoglobulins specific features characterized by immunoglobulin fragments variable (Fv) region, i.e. VH and/or VL
- C07K2317/565—Complementarity determining region [CDR]
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/76—Antagonist effect on antigen, e.g. neutralization or inhibition of binding
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/90—Immunoglobulins specific features characterized by (pharmaco)kinetic aspects or by stability of the immunoglobulin
- C07K2317/92—Affinity (KD), association rate (Ka), dissociation rate (Kd) or EC50 value
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/90—Immunoglobulins specific features characterized by (pharmaco)kinetic aspects or by stability of the immunoglobulin
- C07K2317/94—Stability, e.g. half-life, pH, temperature or enzyme-resistance
Abstract
본 발명은 플라스미노겐 활성인자 저해제-1(PAI-1)에 특이적으로 결합하는 항체를 제공한다. 또한, 본 발명은 항-PAI-1 항체를 암호화하는 핵산, 이러한 항체 또는 이의 단편을 제조하기 위한 재조합 발현 벡터 및 숙주 세포뿐 아니라 약학적 조성물을 제공한다. 시험관 내 또는 생체 내에서 PAI-1 활성을 조절하거나 PAI-1을 검출하기 위해 항체를 사용하는 방법도 또한 제공된다. 본 개시는 또한 활성형 상태에서 PAI-1에 특이적으로 결합하는 항체를 제조하기 위한 방법을 제공한다.
Description
본 출원은 2013년 8월 13일에 출원된 미국 가출원 번호 61/865,451, 및 2014년 5월 22일에 출원된 유럽 특허 출원 번호 14305757.8의 우선권을 주장하며, 이들은 그 전체가 본원에 참조로서 포함된다.
플라스미노겐 활성인자 저해제-1형(plasminogen activator inhibitor type-1, PAI-1)은 플리스미노겐 형성을 담당하는 핵심 세린 프로테아제인 조직형 플라스미노겐 활성인자(tPA) 및 유로키나제형 플라스미노겐 활성인자(uPA)의 주요한 저해제이다. PAI-1은 혈관 구역에서 플라스미노겐 활성화를 저해함으로써 섬유소 용해를 조절한다. 섬유소 용해는 응고 캐스케이드의 활성화에 의해 형성된 섬유소 응괴를 분해하기 위한 엄격하게 조직화된 과정이다. 응고/섬유소 용해 균형의 조절장애는 출혈 또는 혈전성 질환과 같은 이상 지혈 사건을 야기한다. 또한, PAI-1은 수용체에 결합된 플라스미노겐이 유로키나제 수용체(uPAR)에 결합된 유로키나제에 의해 주로 활성화되는 세포주위 구역(혈관 및 조직) 내 플라스미노겐 활성화의 핵심 조절인자이다. 세포주위 단백질 가수분해를 저해함으로써, PAI-1은 세포외 기질(ECM) 분해, 성장 인자 활성화 및 ECM으로부터의 방출, 매트릭스 메탈로프로테이나제(matrix metalloproteinases, MMP) 활성화 및 세포 아폽토시스와 같은 다수의 세포 기능을 조절한다. 최근, (비트로넥틴, 헤파린, 글리코사미노글리칸과 같은) 보조인자, uPAR-유로키나제 복합체 또는 세포 수용체(LRP: 저밀도 지단백질 수용체 관련 단백질), 또는 부착/탈부착, 이동, 증식 및 세포내 생물활성과 같은 세포 기능에 영향을 미치는 인테그린과의 상호작용을 통한 프로테아제에 독립적인 PAI-1의 효과가 밝혀졌다. 이러한 세포 기전 및 항 섬유소 용해 효과에 의해, 종양 생장 및 전이, 섬유증, 급성 심근 경색 및(죽상동맥경화증, 비만 및 당뇨병과 같은) 대사 장애에서의 PAI-1의 병원성 역할이 확립된다.
인간 SERPINE1(PAI-1) 유전자는 8개의 인트론 및 9개의 엑손으로 이루어진 염색체 7에 위치하고 12,169 b의 크기를 가진다(Klinger, K.W. et al. Proc . Natl . Acad. Sci. USA 84:8548, 1987). PAI-1은 세린 프로테아제 저해제(serine protease inhibitor, SERPIN) 슈퍼패밀리로부터의 대략 50 kDa(379개 아미노산)의 단쇄 당단백질로서 활성 형태로 합성되지만 비트로넥틴(Vn)의 부재 시 자발적으로 잠복성이 된다. PAI-1의 주요 보조인자인 비트로넥틴은 표면에 노출된 대략 20개의 아미노산인 반응성 중심 루프(RCL)로 활성 형태를 안정화한다. PAI-1의 2가지 주요한 표적(tPA 및 uPA)의 저해 기전은 자살 저해다. PAI-1의 RCL 영역은 이러한 세린 프로테아제에 대한 절단 자리를 지니고 있는 미끼 펩티드 결합(R346-M347, 또한 P1-P'1로도 불림)을 지닌다. tPA 또는 uPA와의 미카엘리스(Michaelis) 복합체가 우선 형성된 다음, 촉매적 트리아드(triad)가 미끼 펩티드 결합과 반응하여 아실 효소 복합체를 형성하고, 이러한 복합체는 P1-P'1 펩티드 결합의 절단 후, 강한 형태 변화를 유도한다. 형태 변화는 PAI-1을 가지는 아실 효소처럼 공유 결합된 채로 유지되는 프로테아제를 가지는 β-가닥 내로의 절단된 RCL의 삽입을 유발한다. 비생리학적 환경 하에, 이러한 아실 효소 복합체의 가수분해는 절단된 PAI-1 및 유리 활성화 프로테아제의 방출을 유도할 수 있다(Blouse et al., Biochemistry, 48:1723, 2009).
PAI-1은 t-PA 또는 uPA 농도를 초과하는 매우 다양한 수준(nM 범위)으로 혈액 내에서 순환한다. PAI-1는 구조적 유연성을 보이며, 다음의 3가지 형태 중 하나로 발견될 수 있다: (1) 잠복성 형태, (2) 활성 형태, 또는 (3) 기질 형태(도 1 참조). PAI-1은 주로 잠복성 이행을 1.5 내지 3배만큼 낮추는 비트로넥틴과의 비공유 복합체(Kd 약 1 nM)로 발견된다. 잠복성이거나 절단되거나 복합체화된 PAI-1의 비트로넥틴에 대한 친화도는 유의하게 감소한다. 기질에 결합된 비트로넥틴도 또한 세포주위 공간에 PAI-1과 함께 국소화된다. 내피 세포, 단핵구, 대식구 및 혈관 평활근 세포가 이러한 PAI-1을 합성하는데, PAI-1은 그런 다음 혈소판(α 과립 내)에 의해 잠복성 형태 하에 다량으로 저장될 수 있다. PAI-1은 용액 내 tPA 및 uPA의 빠르고 특이적인 저해제(106 내지 107 M-1s-1의 2차 속도 상수를 가짐)이지만, 섬유소 또는 섬유소의 세포 수용체에 결합한 프로테아제에 대해 불활성이다. 덜 효율적이지만, 트롬빈, 플라스민, 활성화 단백질 C와 같은 다른 프로테아제들도 또한 PAI-1에 의해 저해될 수 있다.
잠복 형태로 첫번째 구조가 1992년에 기술된 이래(Mottonen et al., Nature 355:270, 1992), 인간 PAI-1의 몇몇 3D 구조가 해결되었다. 이러한 3D 구조는 기질 내 PAI-1의 돌연변이 형태(Aertgeerts et al., Proteins 23:118, 1995), 안정화된 활성 형태(Sharp et al., Structure 7:111, 1999), 비트로넥틴-소마토메딘 B 도메인에 복합체화된 PAI(Zhou et al., Nat. Struct . Biol. 10:541, 2003) 또는 RCL 루프로부터의 저해 펜타펩티드를 가지는 PAI(Xue et al., Structure 6:627, 1998)를 포함한다. 보다 최근에는, 잠복성 형태인 마우스 PAI-1 구조가 Dewilde 등(J Struct. Biol. 171:95, 2010)에 의해 규명되었고, RCL 위치, 게이트 영역 및 α-나선 A의 위치에서 인간 PAI-1과의 차이가 밝혀졌다. PAI-1에서의 구조/기능 관계를 이러한 다기능 세르핀의 다양한 활성에 관여된 도메인을 국소화하기 위해 600개를 초과하는 돌연변이 단백질(De Taeye et al., Thromb . Haemost. 92:898, 2004에 의해 검토됨)을 이용하여 연구하였다.
PAI-1은 간세포, 지방세포, 혈관사이세포, 섬유아세포, 근섬유아세포, 및 상피 세포를 포함하는 거의 모든 세포 유형에 의해 합성될 수 있기 때문에, PAI-1의 발현은 생리학적(예를 들어, 혈장 PAI-1 수준의 주기적 변동) 및 병리학적 조건(예를 들어, 비만, 대사 증후군, 인슐린 저항성, 감염, 염증성 질환, 암) 하에 매우 다르다. PAI-1은 급성기 단백질로 간주된다. PAI-1 mRNA 발현의 전사 조절은 몇몇 사이토카인 및 성장 인자(예를 들어, TGFβ, TNFα, EGF, FGF, 인슐린, 안지오텐신 II & IV), 호르몬(예를 들어, 알도스테론, 글루코코르티코이드, PMA, 고글루코스) 및 스트레스 인자(예를 들어, 저산소증, 반응성 산소종, 지질다당류)에 의해 유도된다.
더욱이, PAI-1 유전자의 프로모터(위치 - 675) 내 다형성이 발현 수준에 영향을 미친다. 4G 대립형질은 PAI-1 수준을 증가키시고 4G/4G 변이체(군집의 25% 정도에서 발생)은 5G/5G(25% 발생 및 4G/5G 50% 발생)에 비하여 혈장 PAI-1 수준의 대략 25%의 증가를 유도한다. 4G/4G 다형성은 심근 경색(Dawson et al., Arterioscler Thromb. 11:183, 1991), 특정 유형의 폐 섬유증(특발성 간질성 폐렴)(Kim et al., Mol Med. 9:52, 2003)에 관련되어 있고 4G/4G 유전형 공여자 군은 간질성 섬유증&관형 위축으로 인한 신장 이식 소실에 대한 독립적인 위험 인자이다(Rerolle et al., Nephrol. Dial. Transplant 23:3325, 2008).
동맥 및 정맥 혈전증, 급성 심근 경색, 및 죽상동맥경화증과 같은 혈전성 질환에서의 몇몇 병원성 역할이 PAI-1에 기인해 왔다. 인슐린 저항성 증후군 및 비만과 같은 대사성 장애에서의 PAI-1의 관련성이 잘 알려져 있다. 또한 PAI-1은 몇몇 기관에 대한 전섬유화 인자(profibrotic factor)로도 알려져 있으며 섬유증 조직(즉, 간, 폐, 신장, 심장, 복부 유착부, 피부: 흉터 또는 경화증)내에서 과발현되는 것으로 보인다(Ghosh and Vaughan, J. Cell Physiol. 227:493, 2012에 의해 검토됨). PAI-1 녹아웃(KO) 마우스는 상이한 모델, 예를 들어, 간(담관 결찰 또는 생체이물), 신장(일측성 요로폐쇄 모델(UUO)), 폐(블레오마이신 흡입)에서 섬유증으로부터 보호되는 반면(Bauman et al., J. Clin . Invest. 120:1950, 2010; Hattori et al., Am. J. Pathol. 164:1091, 2004; Chuang-Tsai et al., Am. J. Pathol .163:445, 2003), 심장에서의 이러한 결실은 유도성 섬유증으로부터는 보호되지만(Takeshita et al., AM. J. Pathol. 164:449, 2004), 노인성 심장 선택성 섬유증에 걸리기 쉽다(Moriwaki et al., Cric . Res. 95:637, 2004). siRNA에 의한 PAI-1 발현의 하향 조절(Senoo et al., Thorax 65:334, 2010) 또는 화학적 화합물에 의한 저해(Izuhara et al., Arterioscler . Thromb. Vasc . Biol. 28:672, 2008; Huang et al., Am. J. Respir . Cell Mol . Biol. 46:87, 2012)는 폐 섬유증을 낮추는 것으로 보고된 반면 야생형의 PAI-1 과발현(Eitzman et al., J. Clin . Invest. 97:232, 1996) 또는 비트로넥틴 결합만을 보유하고 tPA 저해 기능은 보유하지 않는 PAI-1 돌연변이는 폐 섬유증을 악화시킨다(Courey et al., Blood 118:2313, 2011).
담관 결찰(BDL) 간 섬유증은 PAI-1을 중화하는 항체에 의해 약화되며(미국 특허 번호 7,771,720), siRNA에 의한 하향 조절은 BDL 및 생체이물 유도 간 섬유증을 약화시킨다(Hu et al., J. Hepatol. 51:102, 2009). PAI-1 KO 마우스는 BDL 내 담즙 울체성 유도 간 손상 및 섬유증(Bergheim et al., J. Pharmacol . Exp . Ther. 316:592, 2006; Wang et al., FEBS Lett. 581:3098, 2007; Wang et al., Hepatology 42:1099, 2005) 및 안지오텐신 II 유도 간 섬유증(Beier et al., Arch. Bioch. Biophys. 510:19, 2011)으로부터 보호되었다.
PAI-1 KO 마우스는 UUO 모델(Oda et al., Kidney Int . 60, 587, 2001), 당뇨 신장병(Nicholas et al., Kidney Int. 67:1297, 2005) 및 안지오텐신 II 유도 신장병(Knier et al., J. Hypertens. 29:1602, 2011; 검토를 위해, Ma et al. Frontiers Biosci. 14:2028, 2009 및 Eddy A.A. Thromb . Haemost. 101:656, 2009를 참조)에서 신장 섬유증으로부터 보호된다. 반대로, PAI-1을 과발현하는 마우스는 UUO 후 보다 심각한 섬유증 및 증가된 대식세포 동원을 보인다(Matsuo et al., Kidney Int. 67: 2221, 2005; Bergheim et al., J. Pharmacol . Exp . Ther . 316:592, 2006). 비저해성 PAI-1 돌연변이(PAI-1 R)는 랫트에서 비뇨기 단백질 발현 및 사구체 기질의 축적을 낮춤으로써, 실험적인 사구체신염(thy1)에서 섬유증의 발달로부터 마우스를 보호하는 것으로 나타났다(Huang et al., Kidney Int. 70:515, 2006). PAI-1를 차단하는 펩티드는 UUO 마우스에서 콜라겐 3, 4 및 피브로넥틴 축적을 저해한다(Gonzalez et al., Exp. Biol. Med. 234:1511, 2009).
다수의 병리에 대한 표적으로서 PAI-1은 지난 20년간 PAI-1의 활성을 저해하거나 PAI-1의 발현을 조절하기 위한 집중적인 연구의 중점이 되어 왔다. 화학적 화합물(Suzuki et al., Expert Opin . Investig . Drugs 20:255, 2011), 단일클론 항체(Gils and Declerk, Thromb Haemost; 91:425, 2004), 펩티드, 돌연변이(Cale and Lawrence, Curr . Drug Targets 8:971, 2007), siRNA 또는 안티센스 RNA가 PAI-1의 다양한 기능을 저해하고 PAI-1의 발현을 조절하도록 설계되어왔다. 그러나, 이러한 집중적인 연구에도 불구하고, 치료적 유효량의 PAI-1의 조절제를 개발하는 과제는 여전히 해결되지 않은 채 남아있다. 따라서, PAI-1-매개 인간 병리의 치료에 사용하기 위해 PAI-1 활성을 저해하는 신규한 제제에 대한 수요가 당해 분야에 여전히 존재한다.
일 양태에서, 인간 플라스미노겐 활성인자 저해제 1형(PAI-1)에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시되되, 항체는 중쇄 가변 영역(상기 중쇄 가변 영역은 서열 번호 6의 CDR1(서열 번호 34), CDR2(서열 번호 33), 및 CDR3(서열 번호 32)을 포함함), 및 경쇄 가변 영역(상기 경쇄 가변 영역은 서열 번호 7의 CDR1(서열 번호 37), CDR2(서열 번호 36), 및 CDR3(서열 번호 35)을 포함함)을 포함한다. 추가적인 일 양태에서 중쇄는 서열 번호 6을 포함하는 중쇄 가변 영역을 포함하고, 경쇄는 서열 번호 7을 포함하는 경쇄 가변 영역을 포함한다. 추가의 일 양태에서, 중쇄 가변 영역은 서열 번호 6에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하고, 경쇄 가변 영역은 서열 번호 7에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하다. 모든 % 동일성 근사치들은 최소 % 동일성을 나타내고; 인용된 값보다 더 높은 % 동일성도 또한 본 개시에 포함된다.
다른 양태에서, (a) 중쇄 구조형성(framework) 영역, 서열 번호 34를 포함하는 중쇄 CDR1 영역, 서열 번호 33을 포함하는 중쇄 CDR2 영역, 및 서열 번호 32를 포함하는 중쇄 CDR3 영역; 및 (b) 경쇄 구조형성 영역, 서열 번호 37을 포함하는 경쇄 CDR1 영역, 서열 번호 36을 포함하는 경쇄 CDR2 영역, 및 서열 번호 35를 포함하는 경쇄 CDR3 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시된다. 일정한 양태에서, 항체 중쇄는 서열 번호 6의 중쇄 구조형성 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일한 중쇄 구조형성 영역을 포함하고, 항체 경쇄는 서열 번호 7의 구조형성 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일한 경쇄 구조형성 영역을 포함한다.
일 양태에서, 인간 플라스미노겐 활성인자 저해제 1형(PAI-1)에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시되되, 항체는 중쇄 가변 영역(상기 중쇄 가변 영역은 서열 번호 2의 CDR1(서열 번호 22), CDR2(서열 번호 21), 및 CDR3(서열 번호 20)을 포함함), 및 경쇄 가변 영역(상기 경쇄 가변 영역은 서열 번호 3의 CDR1(서열 번호 25), CDR2(서열 번호 24), 및 CDR3(서열 번호 23)을 포함함)을 포함한다. 추가적인 일 양태에서 중쇄는 서열 번호 2를 포함하는 중쇄 가변 영역을 포함하고, 경쇄는 서열 번호 3을 포함하는 경쇄 가변 영역을 포함한다. 추가의 일 양태에서, 중쇄 가변 영역은 서열 번호 2에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하고, 경쇄 가변 영역은 서열 번호 3에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하다.
추가적인 일 양태에서, (a) 중쇄 구조형성 영역, 서열 번호 22를 포함하는 중쇄 CDR1 영역, 서열 번호 21을 포함하는 중쇄 CDR2 영역, 및 서열 번호 20을 포함하는 중쇄 CDR3 영역; 및 (b) 경쇄 구조형성 영역, 서열 번호 25를 포함하는 경쇄 CDR1 영역, 서열 번호 24를 포함하는 경쇄 CDR2 영역, 및 서열 번호 23을 포함하는 경쇄 CDR3 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시된다. 일정한 양태에서, 항체 중쇄는 서열 번호 26의 중쇄 구조형성 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일한 중쇄 구조형성 영역을 포함하고, 항체 경쇄는 서열 번호 3의 구조형성 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일한 경쇄 구조형성 영역을 포함한다.
일 양태에서, 인간 플라스미노겐 활성인자 저해제 1형(PAI-1)에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시되되, 항체는 중쇄 가변 영역(상기 중쇄 가변 영역은 서열 번호 4의 CDR1(서열 번호 28), CDR2(서열 번호 27), 및 CDR3(서열 번호 26)을 포함함), 및 경쇄 가변 영역(상기 경쇄 가변 영역은 서열 번호 5의 CDR1(서열 번호 31), CDR2(서열 번호 30), 및 CDR3(서열 번호 29)을 포함함)을 포함한다. 추가적인 일 양태에서, 중쇄는 서열 번호 4를 포함하는 중쇄 가변 영역을 포함하고, 경쇄는 서열 번호 5를 포함하는 경쇄 가변 영역을 포함한다. 추가의 일 양태에서, 중쇄 가변 영역은 서열 번호 4에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하고, 경쇄 가변 영역은 서열 번호 5에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하다.
추가적인 일 양태에서, (a) 중쇄 구조형성 영역, 서열 번호 28을 포함하는 중쇄 CDR1 영역, 서열 번호 27을 포함하는 중쇄 CDR2 영역, 및 서열 번호 26을 포함하는 중쇄 CDR3 영역; 및 (b) 경쇄 구조형성 영역, 서열 번호 31을 포함하는 경쇄 CDR1 영역, 서열 번호 30을 포함하는 경쇄 CDR2 영역, 및 서열 번호 29를 포함하는 경쇄 CDR3 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시된다. 일정한 양태에서, 항체 중쇄는 서열 번호 4의 중쇄 구조형성 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일한 중쇄 구조형성 영역을 포함하고, 항체 경쇄는 서열 번호 5의 구조형성 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일한 경쇄 구조형성 영역을 포함한다.
일 양태에서, 인간 플라스미노겐 활성인자 저해제 1형(PAI-1)에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시되되, 항체는 중쇄 가변 영역(상기 중쇄 가변 영역은 서열 번호 8의 CDR1(서열 번호 40), CDR2(서열 번호 39), 및 CDR3(서열 번호 38)을 포함함), 및 경쇄 가변 영역(상기 경쇄 가변 영역은 서열 번호 9의 CDR1(서열 번호 43), CDR2(서열 번호 42), 및 CDR3(서열 번호 41)을 포함함)을 포함한다. 추가적인 일 양태에서 중쇄는 서열 번호 8을 포함하는 중쇄 가변 영역을 포함하고, 경쇄는 서열 번호 9를 포함하는 경쇄 가변 영역을 포함한다. 추가의 일 양태에서, 중쇄 가변 영역은 서열 번호 8에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하고, 경쇄 가변 영역은 서열 번호 9에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하다.
다른 양태에서, (a) 중쇄 구조형성 영역, 서열 번호 40을 포함하는 중쇄 CDR1 영역, 서열 번호 39를 포함하는 중쇄 CDR2 영역, 및 서열 번호 38을 포함하는 중쇄 CDR3 영역; 및 (b) 경쇄 구조형성 영역, 서열 번호 43을 포함하는 경쇄 CDR1 영역, 서열 번호 42를 포함하는 경쇄 CDR2 영역, 및 서열 번호 41을 포함하는 경쇄 CDR3 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시된다. 일정한 양태에서, 항체 중쇄는 서열 번호 8의 중쇄 구조형성 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일한 중쇄 구조형성 영역을 포함하고, 항체 경쇄는 서열 번호 9의 구조형성 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일한 경쇄 구조형성 영역을 포함한다.
일 양태에서, 인간 플라스미노겐 활성인자 저해제 1형(PAI-1)에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시되되, 항체는 중쇄 가변 영역(상기 중쇄 가변 영역은 서열 번호 10의 CDR1(서열 번호 52), CDR2(서열 번호 51), 및 CDR3(서열 번호 50)을 포함함), 및 경쇄 가변 영역(상기 경쇄 가변 영역은 서열 번호 11의 CDR1(서열 번호 55), CDR2(서열 번호 54), 및 CDR3(서열 번호 53)을 포함함)을 포함한다. 추가적인 일 양태에서 중쇄는 서열 번호 10을 포함하는 중쇄 가변 영역을 포함하고, 경쇄는 서열 번호 11을 포함하는 경쇄 가변 영역을 포함한다. 추가의 일 양태에서, 중쇄 가변 영역은 서열 번호 10에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하고, 경쇄 가변 영역은 서열 번호 11에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하다.
추가적인 일 양태에서, (a) 중쇄 구조형성 영역, 서열 번호 52를 포함하는 중쇄 CDR1 영역, 서열 번호 51을 포함하는 중쇄 CDR2 영역, 및 서열 번호 50을 포함하는 중쇄 CDR3 영역; 및 (b) 경쇄 구조형성 영역, 서열 번호 55를 포함하는 경쇄 CDR1 영역, 서열 번호 54를 포함하는 경쇄 CDR2 영역, 및 서열 번호 53을 포함하는 경쇄 CDR3 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시된다. 일정한 양태에서, 항체 중쇄는 서열 번호 10의 중쇄 구조형성 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일한 중쇄 구조형성 영역을 포함하고, 항체 경쇄는 서열 번호 11의 구조형성 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일한 경쇄 구조형성 영역을 포함한다.
일 양태에서, 인간 플라스미노겐 활성인자 저해제 1형(PAI-1)에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시되되, 항체는 중쇄 가변 영역(상기 중쇄 가변 영역은 서열 번호 12의 CDR1(서열 번호 58), CDR2(서열 번호 57), 및 CDR3(서열 번호 56)을 포함함), 및 경쇄 가변 영역(상기 경쇄 가변 영역은 서열 번호 13의 CDR1(서열 번호 61), CDR2(서열 번호 60), 및 CDR3(서열 번호 59)을 포함함)을 포함한다. 추가적인 일 양태에서 중쇄는 서열 번호 12를 포함하는 중쇄 가변 영역을 포함하고, 경쇄는 서열 번호 13을 포함하는 경쇄 가변 영역을 포함한다. 추가의 일 양태에서, 중쇄 가변 영역은 서열 번호 12에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하고, 경쇄 가변 영역은 서열 번호 13에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하다.
다른 양태에서, (a) 중쇄 구조형성 영역, 서열 번호 58을 포함하는 중쇄 CDR1 영역, 서열 번호 57을 포함하는 중쇄 CDR2 영역, 및 서열 번호 56을 포함하는 중쇄 CDR3 영역; 및 (b) 경쇄 구조형성 영역, 서열 번호 61을 포함하는 경쇄 CDR1 영역, 서열 번호 60을 포함하는 경쇄 CDR2 영역, 및 서열 번호 59를 포함하는 경쇄 CDR3 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시된다. 일정한 양태에서, 항체 중쇄는 서열 번호 12의 중쇄 구조형성 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일한 중쇄 구조형성 영역을 포함하고, 항체 경쇄는 서열 번호 13의 구조형성 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일한 경쇄 구조형성 영역을 포함한다.
일 양태에서, 인간 플라스미노겐 활성인자 저해제 1형(PAI-1)에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시되되, 항체는 중쇄 가변 영역(상기 중쇄 가변 영역은 서열 번호 14의 CDR1(서열 번호 64), CDR2(서열 번호 63), 및 CDR3(서열 번호 62)을 포함함), 및 경쇄 가변 영역(상기 경쇄 가변 영역은 서열 번호 15의 CDR1(서열 번호 67), CDR2(서열 번호 66), 및 CDR3(서열 번호 65)을 포함함)을 포함한다. 추가적인 일 양태에서 중쇄는 서열 번호 14의 중쇄 가변 영역을 포함하고, 경쇄는 서열 번호 15의 경쇄 가변 영역을 포함한다. 추가의 일 양태에서, 중쇄 가변 영역은 서열 번호 14에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하고, 경쇄 가변 영역은 서열 번호 15에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하다.
추가적인 일 양태에서, (a) 중쇄 구조형성 영역, 서열 번호 64를 포함하는 중쇄 CDR1 영역, 서열 번호 63을 포함하는 중쇄 CDR2 영역, 및 서열 번호 62를 포함하는 중쇄 CDR3 영역; 및 (b) 경쇄 구조형성 영역, 서열 번호 67을 포함하는 경쇄 CDR1 영역, 서열 번호 66을 포함하는 경쇄 CDR2 영역, 및 서열 번호 65를 포함하는 경쇄 CDR3 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시된다. 일정한 양태에서, 항체 중쇄는 서열 번호 14의 중쇄 구조형성 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일한 중쇄 구조형성 영역을 포함하고, 항체 경쇄는 서열 번호 15의 구조형성 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일한 경쇄 구조형성 영역을 포함한다.
일 양태에서, 인간 플라스미노겐 활성인자 저해제 1형(PAI-1)에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시되되, 항체는 중쇄 가변 영역(상기 중쇄 가변 영역은 서열 번호 16의 CDR1(서열 번호 70), CDR2(서열 번호 69), 및 CDR3(서열 번호 68)을 포함함), 및 경쇄 가변 영역(상기 경쇄 가변 영역은 서열 번호 17의 CDR1(서열 번호 73), CDR2(서열 번호 72), 및 CDR3(서열 번호 71)을 포함함)을 포함한다.
추가적인 일 양태에서 중쇄는 서열 번호 16을 포함하는 중쇄 가변 영역을 포함하고, 경쇄는 서열 번호 17을 포함하는 경쇄 가변 영역을 포함한다. 추가의 일 양태에서, 중쇄 가변 영역은 서열 번호 16에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하고, 경쇄 가변 영역은 서열 번호 17에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하다.
추가적인 일 양태에서, (a) 중쇄 구조형성 영역, 서열 번호 70을 포함하는 중쇄 CDR1 영역, 서열 번호 69를 포함하는 중쇄 CDR2 영역, 및 서열 번호 68을 포함하는 중쇄 CDR3 영역; 및 (b) 경쇄 구조형성 영역, 서열 번호 73을 포함하는 경쇄 CDR1 영역, 서열 번호 72를 포함하는 경쇄 CDR2 영역, 및 서열 번호 71을 포함하는 경쇄 CDR3 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시된다. 일정한 양태에서, 항체 중쇄는 서열 번호 16의 중쇄 구조형성 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일한 중쇄 구조형성 영역을 포함하고, 항체 경쇄는 서열 번호 17의 구조형성 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일한 경쇄 구조형성 영역을 포함한다.
일 양태에서, 인간 플라스미노겐 활성인자 저해제 1형(PAI-1)에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시되되, 항체는 중쇄 가변 영역(상기 중쇄 가변 영역은 서열 번호 80의 CDR1(서열 번호 46), CDR2(서열 번호 45), 및 CDR3(서열 번호 44)을 포함함), 및 경쇄 가변 영역(상기 경쇄 가변 영역은 서열 번호 81의 CDR1(서열 번호 49), CDR2(서열 번호 48), 및 CDR3(서열 번호 47)을 포함함)을 포함한다.
추가적인 일 양태에서 중쇄는 서열 번호 80을 포함하는 중쇄 가변 영역을 포함하고, 경쇄는 서열 번호 81을 포함하는 경쇄 가변 영역을 포함한다. 추가의 일 양태에서, 중쇄 가변 영역은 서열 번호 80에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하고, 경쇄 가변 영역은 서열 번호 81에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하다.
일 양태에서, (a) 중쇄 구조형성 영역, 서열 번호 46을 포함하는 중쇄 CDR1 영역, 서열 번호 45를 포함하는 중쇄 CDR2 영역, 및 서열 번호 44를 포함하는 중쇄 CDR3 영역; 및 (b) 경쇄 구조형성 영역, 서열 번호 49를 포함하는 경쇄 CDR1 영역, 서열 번호 48을 포함하는 경쇄 CDR2 영역, 및 서열 번호 47을 포함하는 경쇄 CDR3 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시된다. 일정한 양태에서, 항체 중쇄는 서열 번호 80의 중쇄 구조형성 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일한 중쇄 구조형성 영역을 포함하고, 항체 경쇄는 서열 번호 81의 구조형성 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일한 경쇄 구조형성 영역을 포함한다.
다른 양태에서, 플라스미노겐 활성인자 저해제 1형(PAI-1)에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시되되, 항체는 중쇄 가변 영역(상기 중쇄 가변 영역은 서열 번호 18의 CDR1(서열 번호 76), CDR2(서열 번호 75), 및 CDR3(서열 번호 74)을 포함함), 및 경쇄 가변 영역(상기 경쇄 가변 영역은 SEQ ID 19의 CDR1(서열 번호 79), CDR2(서열 번호 78), 및 CDR3(서열 번호 77)을 포함함)을 포함한다. 추가적인 일 양태에서 중쇄는 서열 번호 18을 포함하는 중쇄 가변 영역을 포함하고, 경쇄는 서열 번호 19를 포함하는 경쇄 가변 영역을 포함한다. 추가의 일 양태에서, 중쇄 가변 영역은 서열 번호 18에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하고, 경쇄 가변 영역은 서열 번호 19에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하다.
(a) 중쇄 구조형성 영역, 서열 번호 76를 포함하는 중쇄 CDR1 영역, 서열 번호 75를 포함하는 중쇄 CDR2 영역, 및 서열 번호 74를 포함하는 중쇄 CDR3 영역; 및 (b) 경쇄 구조형성 영역, 서열 번호 79를 포함하는 경쇄 CDR1 영역, 서열 번호 78을 포함하는 경쇄 CDR2 영역, 및 서열 번호 77을 포함하는 경쇄 CDR3 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체. 일정한 양태에서, 항체 중쇄는 서열 번호 18의 중쇄 구조형성 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일한 중쇄 구조형성 영역을 포함하고, 항체 경쇄는 서열 번호 19의 구조형성 영역의 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일한 경쇄 구조형성 영역을 포함한다.
일 양태에서, (a) 중쇄 구조형성 영역, 서열 번호 33을 포함하는 중쇄 CDR1 영역, 서열 번호 146을 포함하는 중쇄 CDR2 영역, 및 서열 번호 32를 포함하는 중쇄 CDR3 영역; 및 (b) 경쇄 구조형성 영역, 서열 번호 37을 포함하는 경쇄 CDR1 영역, 서열 번호 145를 포함하는 경쇄 CDR2 영역, 및 서열 번호 35를 포함하는 경쇄 CDR3 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시된다.
일 양태에서, (a) 중쇄 구조형성 영역, 서열 번호 147을 포함하는 중쇄 CDR1 영역, 서열 번호 33을 포함하는 중쇄 CDR2 영역, 및 서열 번호 32를 포함하는 중쇄 CDR3 영역; 및 (b) 경쇄 구조형성 영역, 서열 번호 37을 포함하는 경쇄 CDR1 영역, 서열 번호 36을 포함하는 경쇄 CDR2 영역, 및 서열 번호 35를 포함하는 경쇄 CDR3 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시된다.
일 양태에서, (a) 중쇄 구조형성 영역, 서열 번호 147을 포함하는 중쇄 CDR1 영역, 서열 번호 33을 포함하는 중쇄 CDR2 영역, 및 서열 번호 32를 포함하는 중쇄 CDR3 영역; 및 (b) 경쇄 구조형성 영역, 서열 번호 37을 포함하는 경쇄 CDR1 영역, 서열 번호 145를 포함하는 경쇄 CDR2 영역, 및 서열 번호 35를 포함하는 경쇄 CDR3 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시된다.
일 양태에서, (a) 중쇄 구조형성 영역, 서열 번호 146을 포함하는 중쇄 CDR1 영역, 서열 번호 33을 포함하는 중쇄 CDR2 영역, 및 서열 번호 32를 포함하는 중쇄 CDR3 영역; 및 (b) 경쇄 구조형성 영역, 서열 번호 37을 포함하는 경쇄 CDR1 영역, 서열 번호 145를 포함하는 경쇄 CDR2 영역, 및 서열 번호 35를 포함하는 경쇄 CDR3 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시된다.
일 양태에서, (a) 중쇄 구조형성 영역, 서열 번호 34를 포함하는 중쇄 CDR1 영역, 서열 번호 33을 포함하는 중쇄 CDR2 영역, 및 서열 번호 32를 포함하는 중쇄 CDR3 영역; 및 (b) 경쇄 구조형성 영역, 서열 번호 37을 포함하는 경쇄 CDR1 영역, 서열 번호 145를 포함하는 경쇄 CDR2 영역, 및 서열 번호 35를 포함하는 경쇄 CDR3 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시된다.
추가적인 일 양태에서, 중쇄 가변 영역(중쇄 가변 영역은 서열 번호 6의 CDR1(서열 번호 34), CDR2(서열 번호 33), 및 CDR3(서열 번호 32)을 포함함), 및 경쇄 가변 영역(경쇄 가변 영역은 서열 번호 7의 CDR1(서열 번호 37), CDR2(서열 번호 36), 및 CDR3(서열 번호 35)을 포함함)을 포함하는, 단리된 단일클론 항체와 PAI-1 상의 본질적으로 동일한 에피토프에 결합하는 단리된 단일클론 항체가 본원에 개시된다.
일정한 일 양태에서, (a) 중쇄 구조형성 영역, 서열 번호 76을 포함하는 중쇄 CDR1 영역, 서열 번호 75를 포함하는 중쇄 CDR2 영역, 및 서열 번호 74를 포함하는 중쇄 CDR3 영역; 및 (b) 경쇄 구조형성 영역, 서열 번호 79를 포함하는 경쇄 CDR1 영역, 서열 번호 78을 포함하는 경쇄 CDR2 영역, 및 서열 번호 77을 포함하는 경쇄 CDR3 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시된다.
일 양태에서, 인간 PAI-1에 특이적으로 결합하는 인간화 단일클론 항체가 본원에 개시되되, 항체는 (a) 서열 번호 82를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 91을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편; (b) 서열 번호 83을 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 92를 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편; (c) 서열 번호 84를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 93을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편; (d) 서열 번호 85를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 91을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편; (e) 서열 번호 85를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 93을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편; (f) 서열 번호 86을 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 94를 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편; (g) 서열 번호 87을 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 95를 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편; (h) 서열 번호 88을 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 96을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편; (i) 서열 번호 89를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 97을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편; (j) 서열 번호 90을 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 98을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편; (k) 서열 번호 86을 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 93을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편; (l) 서열 번호 86을 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 95를 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편; (m) 서열 번호 89를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 93을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편; 또는 (n) 서열 번호 89를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 95를 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편을 포함한다. 추가의 일 양태에서, 인간화 중쇄 가변 영역은 이전에 개시된 인간 중쇄 가변 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하고, 인간화 경쇄 가변 영역은 이전에 개시된 임의의 인간 경쇄 가변 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하다.
일 양태에서, (a) 중쇄 구조형성 영역 및 서열 번호 86을 포함하는 중쇄 가변 영역, 및 (b) 경쇄구조형성 영역 및 서열 번호 93을 포함하는 경쇄 가변 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시된다. 일정한 양태에서, 단리된 단일클론 중쇄는 서열 번호 86의 중쇄 구조형성 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일한 중쇄 구조형성 영역을 포함하고, 단리된 단일클론 항체 경쇄는 서열 번호 93의 구조형성 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일한 경쇄 구조형성 영역을 포함한다. 일정한 다른 양태에서, 단리된 단일클론 항체 중쇄는 서열 번호 86의 중쇄 구조형성 영에 95% 동일한 중쇄 구조형성 영역을 포함하고, 단리된 단일클론 항체 경쇄는 서열 번호 93의 구조형성 영역에 95% 동일한 경쇄 구조형성 영역을 포함한다.
다른 양태에서, 인간 PAI-1에 특이적으로 결합하는 인간화 단일클론 항체가 본원에 개시되되, 항체는 서열 번호 154를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편; 및 서열 번호 153을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편을 포함한다. 다른 양태에서, 인간 PAI-1에 특이적으로 결합하는 인간화 단일클론 항체가 본원에 개시되되, 항체는 서열 번호 155를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 153을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편을 포함한다. 추가의 일 양태에서, 인간화 중쇄 가변 영역은 이전에 개시된 임의의 인간 중쇄 가변 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하고, 인간화 경쇄 가변 영역은 이전에 개시된 임의의 인간 경쇄 가변 영역에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하다.
다른 양태에서, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시되되, 항체는 서열 번호 158을 포함하는 폴리펩티드에 결합한다. 다른 구현예에서, 단리 단일클론 항체는 서열 번호 158을 포함하는 폴리펩티드의 단편에 결합한다. 또 다른 구현예에서, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체는 서열 번호 156 및/또는 서열 번호 158을 포함하는 폴리펩티드에 결합한다. 다른 구현예에서, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체는 서열 번호 156, 서열 번호 158, 및/또는 서열 번호 157을 포함하는 폴리펩티드에 결합한다. 여전히 다른 구현예에서, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체는 서열 번호 1의 잔기 160, 262, 296~297, 300~307, 및/또는 310~316에 대하여 특이적 결합 친화도를 가진다. 일정한 구현예에서, 본원에 개시된 단리된 단일클론 항체는 적어도 서열 번호 1의 잔기 311, 312, 및 313(D-Q-E)과 상호작용한다. 일정한 구현예에서, 항체에 의해 결합된 PAI-1은 인간 PAI-1이다. 다른 구현예에서, 항체에 의해 결합된 PAI-1은 인간 PAI-1의 활성 형태이다.
다른 구현예에서, 본원에 개시된 PAI-1에 특이적으로 결합하는 단리된 단일클론 항체는 서열 번호 161을 포함하는 폴리펩티드에 결합한다. 여전히 다른 구현예에서, 단리된 단일클론 항체는 서열 번호 159 및/또는 서열 번호 161을 포함하는 폴리펩티드에 결합한다. 여전히 다른 구현예에서, 단리된 단일클론 항체는 서열 번호 159, 서열 번호 160, 및/또는 서열 번호 161을 포함하는 폴리펩티드에 결합한다. 여전히 다른 구현예에서, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체는 시아노-PAI-1(서열 번호 162)의 잔기 44~64 및/또는 잔기 307~321에 대하여 특이적 결합 친화도를 가진다. 일정한 구현예에서, 항체에 의해 결합된 PAI-1는 시아노-PAI-1이다. 다른 구현예에서, 항체에 의해 결합된 PAI-1은 시아노-PAI-1의 잠복성 형태이다.
추가의 일 양태에서, PAI-1에 대한 임의의 개시된 항체의 결합을 경쟁적으로 저해하는 단리된 단일클론 항체가 본원에 개시된다. 일 구현예에서, 본원에 개시된 임의의 단리된 단일클론 항체의 결합을 경쟁적으로 저해하고/하거나 결합에 대해 경쟁하는 단리된 단일클론 항체가 본원에 개시된다. 일정한 구현예에서, 단리된 단일클론 항체는 인간 PAI-1에 대한 결합을 경쟁적으로 저해하거나 경쟁한다. 일정한 구현예에서, 단리된 단일클론 항체는 서열 번호 156, 서열 번호 157, 및/또는 서열 번호 158을 포함하는 폴리펩티드에 대한 결합을 경쟁적으로 저해하거나 경쟁한다. 다른 구현예에서, 단리된 단일클론 항체는 서열 번호 159, 서열 번호 160, 및/또는 서열 번호 161을 포함하는 폴리펩티드에 대한 결합을 경쟁적으로 저해하거나 경쟁한다. 일 구현예에서, 단리된 항체는 (a) 중쇄 구조형성 영역, 서열 번호 34를 포함하는 중쇄 CDR1 영역, 서열 번호 33을 포함하는 중쇄 CDR2 영역, 및 서열 번호 32를 포함하는 중쇄 CDR3 영역; 및 (b) 경쇄 구조형성 영역, 서열 번호 37을 포함하는 경쇄 CDR1 영역, 서열 번호 145를 포함하는 경쇄 CDR2 영역, 및 서열 번호 35를 포함하는 경쇄 CDR3 영역을 포함하는 단리된 단일클론 항체와 서열 번호 156, 157, 및/또는 158을 포함하는 폴리펩티드에 대한 결합에 대해 경쟁한다.
다른 양태에서, 본원에 개시된 임의의 단리된 단일클론 항체를 암호화하는 뉴클레오티드가 본원에 개시된다.
일 양태에서, 환자 또는 다른 대상체에게 약학적 유효량의 PAI-1 항체를 경구로, 주사용 용액에 의해 비경구로, 흡입으로 또는 국소로 투여하는 단계를 포함하는, PAI-1의 증가된 발현 또는 PAI-1에 대한 증가된 감수성으로 인해 유발되는 병태를 치료하는 방법이 본원에 개시된다.
일 양태에서, 필요로 하는 환자 또는 다른 대상체에게 약학적 유효량의 PAI-1 항체를 경구로, 주사용 용액에 의해 비경구로, 흡입으로 또는 국소로 투여하는 단계를 포함하는, 플라스민 형성을 화복하는 방법이 본원에 개시된다. 본원에 개시된 비경구 투여는 정맥 내, 점적, 동맥 내, 복강 내, 근육 내, 피하, 직장 또는 질, 비경구 투여의 정맥 내, 동맥 내, 피하, 및 근육 내 형태를 포함한다. 일부 구현예에서, 환자 또는 다른 대상체에 대한 투여는 다중 투여를 포함한다. 다른 양태에서, 플라스민 형성을 회복하는 방법은 증가된 수준의 섬유증 조직을 포함하는 병태의 치료적 처치를 용이하게 한다. 일부 양태에서, 병태는 섬유증을 특징으로 한다. 일부 양태에서, 병태는 섬유증, 피부 섬유증, 전신성 경화증, 폐 섬유증, 특발성 폐 섬유증, 간질성 폐 질환, 및 만성 폐 질환이다. 다른 양태에서, 플라스민 형성은 간 섬유증, 만성 신장 질환을 포함하는 신장 섬유증, 혈전증, 정맥 및 동맥 혈전증, 심부 정맥혈전증, 말초 사지 허혈, 파종혈관내응고 혈전증, 혈전 용해를 동반하는 그리고 동반하지 않는 급성 허혈성 뇌졸중 또는 스텐트 재협착의 치료적 처치를 용이하게 한다.
다른 양태에서, 환자 또는 다른 대상체에게 경구로, 주사용 용액에 의해 비경구로, 흡입으로 또는 국소로 투여하는 단계를 포함하는, PAI-1의 증가된 발현 또는 PAI-1에 대한 증가된 감수성에 의해 유발되는 병태를 치료하기 위한 약제의 제조를 위한 약학적 유효량의 PAI-1 항체의 용도가 본원에 개시된다.
일 양태에서, 약제는 증가된 수준의 섬유증 조직을 포함하는 병태를 치료하기 위한 것이다. 일부 양태에서, 병태는 섬유증을 특징으로 한다. 일부 양태에서, 병태는 섬유증, 피부 섬유증, 전신성 경화증, 폐 섬유증, 특발성 폐 섬유증, 간질성 폐 질환, 및 만성 폐 질환이다. 다른 양태에서, 약제는 간 섬유증, 만성 신장 질환을 포함하는 신장 섬유증, 혈전증, 정맥 및 동맥 혈전증, 심부 정맥혈전증, 말초 사지 허혈, 파종혈관내응고 혈전증, 혈전 용해를 동반하는 그리고 혈전 용해를 동반하지 않는 급성 허혈성 뇌졸중, 또는 스텐트 재협착을 포함하는 병태를 치료하기 위한 것이다.
다른 양태에서, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시되되, 항체가 폐 섬유증을 저해한다. 일정한 구현예에서, 항체는 대상체의 폐에서 섬유증을 저해한다. 일정한 구현예에서, 항체는 특발성 폐 섬유증(IPF)을 가지는 대상체의 폐에서 섬유증을 저해한다. 일부 구현예에서, 본원에 개시된 단리된 단일클론 항체는 대상체에서 섬유소 분해의 증가를 유도한다. 일정한 구현예에서, 항체는 대상체의 혈장에서 섬유소 분해를 증가시킨다. 일부 다른 구현예에서, 본원에 개시된 단리된 단일클론 항체는 대상체의 폐에서 콜라겐 축적을 저해한다. 일부 구현예에서, 대상체는 IPF를 가진다. 일부 다른 구현예에서, 본원에 개시된 단리된 단일클론 항체는 대상체의 기관지 폐포 세척액(bronchoalveolar lavage fluid, BALF)에서 D-이량체 수준을 증가시킨다. 일부 구현예에서, 대상체는 IPF를 가진다. 일부 다른 구현예에서, 본원에 개시된 단리된 단일클론 항체는 PAI-1에 특이적으로 결합하되, 항체는 대상체에서 섬유증으로 인한 폐 중량의 증가를 저해한다. 일 구현예에서, 대상체는 IPF를 가진다.
다른 양태에서, 환자에게 경구로, 주사용 용액에 의해 비경구로, 흡입으로 또는 국소로 투여하는 단계를 포함하는, PAI-1에 대한 증가된 감수성 또는 PAI-1의 증가된 발현에 의해 유발되는 병태를 치료하기 위한 약제의 제조를 위한 약학적 유효량의 PAI-1 항체의 용도가 본원에 개시되되, 병태는 특발성 폐 섬유증이다.
다른 양태에서, 환자 또는 이의 다른 대상체에게 경구로, 주사용 용액에 의해 비경구로, 흡입으로 또는 국소로 약학적 유효량의 PAI-1 항체를 투여하는 단계를 포함하는, 플라스민 형성을 회복하는 방법이 본원에 개시되되, 플라스민 형성은 특발성 폐 섬유증의 치료적 처치를 용이하게 한다.
다른 양태에서, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시되되, 항체가 대상체에서 섬유소 용해 활성을 회복한다. 일정한 구현예에서, 항체는 급성 허혈성 뇌졸중을 가지는 대상체에서 섬유소 용해 활성을 회복한다. 급성 허혈성 뇌졸중은 혈전 용해를 동반하거나 동반하지 않을 수 있다. 일부 구현예에서, 단리된 단일클론 항체는 응괴 용해를 회복한다. 일정한 구현예에서, 항체는 시험관 내 응괴 용해를 회복한다. 여전히 다른 구현예에서, 항체는 약 2 nM의 IC50으로 시험관 내 응괴 용해를 회복한다.
다른 양태에서, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시되되, 항체는 대상체 내에서 섬유소 분해를 회복한다. 일부 구현예에서, 대상체는 급성 허혈성 뇌졸중을 가진다.
다른 양태에서, 환자에게 경구로, 주사용 용액에 의해 비경구로, 흡입으로 또는 국소로 투여하는 단계를 포함하는, PAI-1에 대한 증가된 감수성 또는 PAI-1의 증가된 발현에 의해 유발되는 병태를 치료하기 위한 약제의 제조를 위한 약학적 유효량의 PAI-1 항체의 용도가 본원에 개시되되, 병태는 혈전 용해를 동반하는 그리고 동반하지 않는 급성 허혈성 뇌졸중이다.
다른 양태에서, 필요로 하는 환자 또는 다른 대상체에게 경구로, 주사용 용액에 의해 비경구로, 흡입으로 또는 국소로 약학적 유효량의 PAI-1 항체를 투여하는 단계를 포함하는, 플라스민 형성을 회복하는 방법이 본원에 개시되되, 플라스민 형성이 혈전 용해를 동반하는 그리고 동반하지 않는 급성 허혈성 뇌졸중의 치료적 처치를 용이하게 한다.
다른 양태에서, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시되되, 항체가 대상체에서 유착의 형성을 저해한다. 일부 구현예에서, 대상체에 대한 유착 형성은 수술 또는 손상 후에 뒤따른다. 일부 구현예에서, 대상체 내 유착 형성은 복부 내이다. 다른 구현예에서, 유착 형성은 대상체의 어깨, 골반, 심장, 척추, 손 및 기타 신체 영역에서 발생한다.
다른 양태에서, 환자에게 경구로, 주사용 용액에 의해 비경구로, 흡입으로 또는 국소로 투여하는 단계를 포함하는, PAI-1에 대한 증가된 감수성 또는 PAI-1의 증가된 발현에 의해 유발되는 병태를 예방하거나 치료하기 위한 약제의 제조를 위한 약학적 유효량의 PAI-1 항체의 용도가 본원에 개시되되, 병태는 복부 유착 형성이다.
다른 양태에서, 필요로 하는 환자 또는 다른 대상체에게 경구로, 주사용 용액에 의해 비경구로, 흡입으로 또는 국소로 약학적 유효량의 PAI-1 항체를 투여하는 단계를 포함하는, 플라스민 형성을 회복하는 방법이 본원에 개시되되, 플라스민 형성은 유착 형성의 예방 또는 치료적 처치를 용이하게 한다. 일부 구현예에서, 대상체 내 유착 형성은 복부이다.
다른 양태에서, PAI-1/비트로넥틴 복합체에 결합하는 단리된 단일클론 항체가 본원에 개시된다. 다른 양태에서, PAI-1 기질 형태를 유도함으로써 PAI-1의 활성을 중화하는 단리된 단일클론 항체가 본원에 개시된다. 일 구현예에서, 항체는 플라스민 형성을 회복시키거나 회복시킬 수 있다. 다른 구현예에서, 단리된 단일클론 항체는 피브로넥틴 분해를 유도하거나 유도할 수 있다. 또 다른 구현예에서, 단리된 단일클론 항체는 매트릭스 메탈로프로테이나제(MMP) 활성화를 유도하거나 유도할 수 있다.
다른 양태에서, 본원에 개시된 단리된 단일클론 항체는 항체 단편이다. 일부 구현예에서, 항체는 단쇄 Fv 항체이다. 다른 구현예에서, 중쇄 및 경쇄는 유연한 링커에 의해 연결되어 단쇄 항체를 형성한다. 다른 구현예에서, 항체는 Fab, Fab', 또는 (Fab')2 항체이다.
다른 양태에서, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체가 본원에 개시되되, 항체는 결정화 항체이다. 일 구현예에서, 단일클론 항체 A44의 Fab' 단편을 포함하는 단리된 결정이 본원에 개시되되, Fab' 단편은 경쇄 서열 서열 번호 7 및 중쇄 서열 서열 번호 6으로 이루어진다. 다른 구현예에서, 경쇄 서열 서열 번호 93 및 중쇄 서열 서열 번호 86을 포함하는 Fab' 단편을 포함하는 단리된 결정이 본원에 개시된다. 일 구현예에서, 단리된 결정은 a=105 Å, b=152 Å 및 c=298 Å의 비대칭 단위 세포 수치를 포함한다. 일 구현예에서, 단리된 결정은 P212121 공간 그룹에 속한다. 다른 구현예에서, 단리된 결정은 3.3 Å 분해능의 x-선 회절을 포함한다. 일 구현예에서, 단리된 결정은 결정화 항체의 생물학적 활성을 보유한다. 일부 구현예에서, 단리된 결정은 결정화 항체의 가용성 대응체보다 큰 생체 내 반감기를 가진다.
일 양태에서, (a) PAI-1에 특이적으로 결합하는 결정화 항체 및 (b) 결정을 내포되거나 캡슐화하는 적어도 하나의 약학적 부형제를 포함하는 약학적 조성물이 본원에 개시된다.
다른 양태에서, 약학적으로 허용가능한 담체 및 치료적 유효량의 본원에 개시된 임의의 항체를 포함하는 약학적 조성물이 본원에 개시된다.
일 양태에서, 포유동물을 PAI-1, 또는 이의 단편, 및 비트로넥틴으로 이루어진 복합체로 면역화하는 단계를 포함하는, PAI-1에 대한 항체를 생성하는 방법이 본원에 개시된다.
다른 양태에서, (a) PAI-1을 ELISA 플레이트에 결합시키는 단계; (b) ELISA 플레이트를 PAI-1 항체와 항온배양하는 단계; (c) ELISA 플레이트를 tPA와 항온배양하는 단계; (d) ELISA 플레이트를 표지된 항-tPA 항체와 항온배양하는 단계; 및 (e) 표지된 항-tPA 항체에 의해 방출되는 OD405를 측정하는 단계를 포함하는, ELISA에서 tPA 활성 저해제로서 PAI-1의 기능을 차단하는 능력에 대하여 PAI-1 항체를 스크리닝하는 방법이 본원에 개시되되, 양성 판독은 PAI-1 항체가 PAI-1에 결합하지만 PAI-1과 tPA 사이의 공유 결합의 형성을 차단하지는 않는다는 것을 나타내고, 음성 판독은 PAI-1항체가 tPA와 PAI-1과의 상호작용을 차단한다는 것을 나타낸다.
다른 양태에서, 하이브리도마를 스크리닝하는 방법이 본원에 개시된다. 일정한 구현예에서, 스크리닝 방법은 항-PAI-1 항체에 고정화된 항-마우스를 이용한 역방향 스크리닝 방법을 포함한다. 다른 구현예에서, 스크리닝 방법은 고정화된 비트로넥틴에 대항하거나 리간드로서 유리 PAI-1을 사용하는 정방향 스크리닝 검정을 포함한다. 일정한 구현예에서, 이러한 방법은 PAI-1/비트로넥틴 복합체에 대한 항체의 친화도를 결정하기 위해 적용된다. 일부 구현예에서, 방법은 비트로넥틴을 표면에 고정하는 단계; PAI-1를 표면에 고정된 비트로넥틴에 접촉시킴으로써 복합체를 형성하는 단계; 복합체를 포함하는 표면을 항체와 접촉시키는 단계; 미결합 항체로부터 복합체에 결합된 항체를 분리하는 단계; 복합체에 결합된 항체를 검출하고, 복합체에 결합된 항체의 수준을 분석하여 복합체에 대한 항체의 친화도를 결정하는 단계를 포함한다.
도 1은 세린 프로테아제 조직형 플라스미노겐 활성인자(tPA) 및 유로키나제형 플라스미노겐 활성인자(uPA)와 PAI-1 사이의 기전의 개략도를 도시한 것이다. PAI-1은 구조적 유연성을 나타내며 비트로넥틴(Vn)에 결합할 때 활성 형태 또는 잠복성 형태로 나타날 수 있다. PAI-1의 RCL 영역은 세린 프로테아제에 의한 절단 자리인 미끼 펩티드 결합(또한, P1-P1'로도 불림)을 지닌다. tPA 또는 uPA와의 미카엘리스(Michaelis) 복합체가 우선 형성된 다음, 촉매적 트리아드(triad)가 미끼 펩티드 결합과 반응하여 아실 효소 복합체를 형성하고, 이러한 복합체는 P1-P'1 펩티드 결합의 절단 후, 강한 형태 변화를 유도한다. 아실 효소는 세린 프로테아제(tPA)로부터의 촉매적 트리아드로부터의 세린 잔기(검은 삼각형)와 기질로부터의 아미노산(검은 원) 사이의 공유 결합에 의해 형성된 불안정한 복합체로 추가의 가수분해를 거친다. 형태 변화는 PAI-1을 가지는 아실 효소처럼 공유 결합된 채로 유지되는 프로테아제로 β-가닥 내로의 절단된 RCL의 삽입을 유발한다. 비생리학적 환경 하에, 이러한 아실 효소 복합체의 가수분해는 절단된 PAI-1 및 유리 활성화 프로테아제의 방출을 유도할 수 있다.
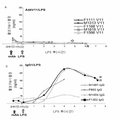
도 2는 실시예 2에 기술된 바와 같은 결합 ELISA에서 항체 역가에 대한 통상적인 표준 곡선을 도시한 것이다. 항체 31C9, 33B8 및 33H1는 양성 대조군이었고 IgG1은 음성 대조군이었다.
도 3은 실시예 4에 기술된 바와 같이 tPA와 PAI-1의 상호작용을 차단하는 선택 항체에 대한 기능적 ELISA에 대한 대표 곡선을 도시한 것이다. 항체 33H1은 양성 대조군이고, IgG1는 음성 대조군이고 A44는 양성 항체 클론으로 확인되었다.
도 4는 실시예 4에 기술된 발색 검정에서 상업적으로 이용가능한 항체(33B8 및 33H1) 및 A44에 의한 tPA의 인간 PAI-1 차단 활성의 중화를 도시한 것이다.
도 5는 상이한 융합으로부터 생성된 항체의 선택에 의한 tPA의 인간 PAI-1 차단 활성의 중화를 도시한 것이다(실시예 4 참조).
도 6은 발색 검정에서 인간 PAI-1 및 그의 오소로그(ortholog)가 인간 tPA 활성을 유사한 효능으로 차단한다는 것을 도시한 것이다.
도 7은 실시예 4에 기술된 발색 검정에서 A44 및 33B8(상업적으로 이용가능함) 항체에 의한 인간 tPA의 시노몰구스(시노) 및 마우스 PAI-1 차단 활성의 중화를 도시한 것이다.
도 8은 항체 33H8(PAI-1을 활성 형태로부터 잠복성 형태로 전환함), 33H1(PAI-1을 활성 형태로부터 기질 형태로 전환함) 및 A44가 PAI-1과 tPA의 상호작용을 차단하는 작용의 기전의 SDS-Page 분석을 도시한 것이다. 레인 1: 분자량 기준; 레인 2: PAI-1 단독; 레인 3: tPA 단독; 레인 4: tPA의 존재 하 PAI-1; 레인 5: 33B8 +PAI-1 +tPA; 레인 6: 33H1+PAI-1+tPA; 레인 7: A44+PAI-1+tPA; 레인 8: mAb는 이소형 대조 항체이다.
도 9는 항체 33H8(PAI-1을 활성 형태로부터 잠복성 형태로 전환함), 33H1(PAI-1을 활성 형태로부터 기질 형태로 전환함) 및 융합 C26, E16 및 E21로부터 개발된 항체의 PAI-1과 tPA의 상호작용을 차단하기 위한 작용의 기전의 SDS-Page 분석을 도시한 것이다. 레인 1: 분자량 기준; 레인 2: PAI-1 단독; 레인 3: tPA 단독; 레인 4: tPA의 존재 하 PAI-1; 레인 5: 33B8 +PAI-1 +tPA; 레인 6: 33H1+PAI-1+tPA; 레인 7: C26+PAI-1+tPA; 레인 8: E16+PAI-1+tPA; 레인 9: E21+PAI-1+tPA; 레인 10: mAb는 이소형 대조 항체이다.
도 10은 항체 33H8(PAI-1을 활성 형태로부터 잠복성 형태로 전환함), 33H1(PAI-1을 활성 형태로부터 기질 형태로 전환함) 및 융합 A39, B109 및 C45로부터 개발된 항체의 PAI-1과 tPA의 상호작용을 차단하기 위한 작용의 기전의 SDS-Page 분석을 도시한 것이다. 레인 1: 분자량 기준; 레인 2: PAI-1 단독; 레인 3: tPA 단독; 레인 4: tPA의 존재 하 PAI-1; 레인 5: 33B8 +PAI-1 +tPA; 레인 6: 33H1+PAI-1+tPA; 레인 7: A39+PAI-1+tPA; 레인 8: B109+PAI-1+tPA; 레인 9: C45+PAI-1+tPA; 레인 10: mAb는 이소형 대조 항체이다.
도 11은 다음의 뮤라인 항체의 경쇄의 정렬을 도시한 것이다: A105(서열 번호 3), A39(서열 번호 5), A44(서열 번호 7), A71(서열 번호 9), A75(서열 번호 81), B109(서열 번호 11), B28(서열 번호 13), C45(서열 번호 15), E16(서열 번호 17), 및 E21(서열 번호 19). CDR은 굵은 글씨로 강조되어 있다.
도 12는 다음의 뮤라인 항체의 중쇄의 정렬을 도시한 것이다: A105(서열 번호 2), A39(서열 번호 4), A44(서열 번호 6), A71(서열 번호 8), A75(서열 번호 80), B109(서열 번호 10), B28(서열 번호 12), C45(서열 번호 14), E16(서열 번호 16), 및 E21(서열 번호 18). IMGT에 의해 정의된 CDR은 굵은 글씨로 강조되어 있다.
도 13은 뮤라인 A44 경쇄(서열 번호 7)와 vk1(서열 번호 101) 및 v람다3(서열 번호 102)의 정렬을 도시한 것이다.
도 14는 뮤라인 A44 중쇄(서열 번호 6)와 vh2(서열 번호 103) 및 vh4(서열 번호 104)의 정렬을 도시한 것이다.
도 15는 클론 A44 인간화 VL과 정렬된 모든 구조체(construct)를 도시한 것이다. 모든 정렬된 서열(서열 번호: 91~98)은 아래의 표 25에 추가로 기술되어 있다. 검은색 상자는 CDR 도메인을 나타낸다. 강조된 잔기는 정렬 내 바로 위의 잔기와 서열이 상이하다. 잔기 번호는 IMGT에 기술된 바와 같다.
도 16은 클론 A44 인간화 VH와 정렬된 모든 구조체를 도시한 것이다. 모든 정렬된 서열(서열 번호: 82~90)은 표 25에 추가로 기술되어 있다. 검은색 상자는 CDR 도메인을 나타낸다. 강조된 잔기는 정렬 내 바로 위의 잔기와 서열이 상이하다. 잔기 번호는 IMGT에 기술된 바와 같다.
도 17은 PAI-1 활성의 저해 백분율이 mAb 농도의 함수로서 도표화되었고, IC50은 Biostat 스피드 소프트웨어를 사용한 Imax로 결정되었다는 것을 도시한다.
도 18은 동종 재조합 6-His 태그 Fab A44의 정제를 도시한 것이다.
도 19는 고정화 APG 항체에 결합된 인간 PAI-1 글리코실화의 단일 운동 분석을 이용한 비아코어 2000으로의 SPR 분석을 도시한 것이다. 단일 주기 운동으로부터의 센서그램은 회색으로 나타낸다. 적합 모델(fit model)은 검은색으로 나타낸다.
도 20은 ELISA에 의한 UK-PAI-1 복합체 형성 검출에 의해 결정된 APG, APGv2, 및 APGv4 항체에 의한 인간 혈장 PAI-1 중화를 도시한 것이다. PAI-1 활성의 저해 백분율은 APG, APGv2, 또는 APGv4 항체의 농도의 함수로서 도표화되었다.
도 21은 시간(분)의 함수로서 340 nm에서의 흡광도 판독에 의한 혼탁 운동 측정으로 검출된 tPA 1 nM 및 PAI-1 3 nM의 존재 하에 A44V11(1, 3 또는 10 nM)에 의한 인간 혈장 응괴 용해의 회복을 도시한 것이다.
도 22는 시간(분)의 함수로서 340 nm에서의 흡광도로 검출된 tPA 1 nM 및 PAI-1 3 nM의 존재 하에 인간 IgG1 음성 대조군(1, 3 또는 10 nM)에 의해서는 인간 혈장 응괴 용해의 회복이 부재함을 도시한 것이다.
도 23은 A44V11 또는 다양한 농도의 인간 IgG1 이소형 음성 대조군에 의한 인간 혈장 응괴 용해의 회복을 도시한 것이다.
도 24는 시간(분)의 함수로 340 nm의 흡광도에서 검출된 tPA 1 nM 및 PAI-1 3 nM의 존재 하에 3 nM에서의 APG, APGV2 또는 APGV4에 의한 인간 혈장 응괴 용해의 회복을 도시한 것이다.
도 25는 다양한 농도의 APG 변이체 2 및 4에 의한 인간 혈장 응괴 용해의 회복을 도시한 것이다.
도 26은 TGFβ 5 ng/ml 및 50 nM의 IgG 이소형 대조군 mAb 또는 A44V11에 의한 처리 48H 후에서의 인간 LL29 근섬유아세포 상층액에 대한 면역블롯 항-PAI-1을 도시한 것이다.
도 27은 PBS(대조군), 플라스미노겐(Pg), A44v11 및 플라스미노겐(A+Pg) 또는 음성 인간 IgG 및 플라스미노겐(Neg+Pg)으로의 48시간 세포 처리 후 인간 일차 폐 섬유아세포 내 전체적인 MMP 활성을 도시한 것이다.
도 28은 4일에 복강 내 투여에 의한 A44 또는 IgG1(10 mg/kg) 또는 PBS로의 처리 후 7일 및 9일에서 블레오마이신 처리된 마우스로부터의 기관지 폐포 세척액(broncho-alveolar lavage fluid, BALF)(a) 및 폐 용해물(b) 내 인간 활성 PAI-1 수준을 도시한 것이다. 활성 PAI-1은 ELISA(# HPAIKT Molecular Innovation)에 의해 결정된다. 저해의 백분율은 A44 블레오와 IgG 블레오 사이의 차를 IgG 블레오 및 미처리(PBS) 마우스 군과의 차로 나눠서 계산하였다.
도 29는 ELISA(Asserachrom D-Di, Diagnostica Stago)에 의해 결정된 4일에복강 내 투여에 의한 A44 또는 IgG1(10 mg/kg) 또는 PBS로의 처리 후 7일 및 9일에서의 블레오마이신 처리된 마우스로부터의 BALF 내 마우스 D-이량체 수준을 도시한 것이다. A44에 의해 유도된 D-이량체 내 배율 증가는 IgG에 비교하여 나타낸다.
도 30은 4일부터 20일까지 3일마다 PBS(비히클), IgG1 또는 A44 10mg/kg 복강 내 투여가 이어지는 염수 또는 블레오마이신 처리 21일 후 유전자이식 인간화 마우스로부터의 우측 폐 중량을 도시한 것이다.
도 31은 4일부터 20일까지 3일마다 PBS(비히클), IgG1 또는 A44 10mg/kg 복강 내 투여가 이어지는 염수 또는 블레오마이신 처리 21일 후 유전자이식 인간화 마우스 내 하이드록시프롤린 폐 함량을 도시한 것이다.
도 32는 LPS 챌린지(100 ug/kg iv) 24시간 전에 A44V11(a) mAb(n=5) 또는 IgG1 이소형 대조군(b)(n=4)(5 mg/kg ip)으로 처리된 원숭이로부터의 혈장 내 활성 PAI-1 수준을 도시한 것이다. 혈액 시료를 지시된 시점에서 수확하였고, 활성 PAI-1 수준을 혈장에서 ELISA(Molecular Innovation으로부터의 # HPAIKT)를 사용하여 결정하였다.
도 33은 LPS 챌린지(100 ug/kg iv) 24시간 전에 A44V11(a) mAb(n=5) 또는 IgG1 이소형 대조군(b)(n=4)(5 mg/kg ip)으로 처리된 원숭이로부터의 간 생검물 내 활성 PAI-1 수준을 도시한 것이다. 간 생검물을 지시된 시점에 마취된 원숭이에서 수확하였고, 용해물 내 활성 PAI-1 수준을 ELISA(Molecular Innovation # HPAIKT)를 사용하여 결정하였다.
도 34는 LPS 챌린지(100 ug/kg iv) 24시간 전에 A44V11(a) mAb(n=5) 또는 IgG1 이소형 대조군(b)(n=4)(5 mg/kg ip)으로 처리된 원숭이로부터의 혈장 내 D-이량체 수준을 도시한 것이다. 혈액 시료를 지시된 시점에서 수확하였고, D-이량체 수준을 혈장에서 ELISA를 사용하여 결정하였다.
도 35는 LPS 챌린지(100 ug/kg iv) 24시간 전에 A44V11(a) mAb(n=5) 또는 IgG1 이소형 대조군(b)(n=4)(5 mg/kg ip)으로 처리된 원숭이로부터의 혈장 내 플라스민-α2 항플라스민(PAP) 복합체 수준을 도시한 것이다. 혈액 시료를 지시된 시점에서 수확하였고, PAP 수준을 혈장에서 ELISA(Diagnostica Stago로부터의 # Asserachrom PAP)를 사용하여 결정하였다.
도 36은 복강 내 유체(intraperitoneal fluid, IPF) 및 자궁각 용해물 내 활성 PAI-1의 수준을 도시한 것이다. 복강 내 유체(intraperitoneal fluid, IPF)(a) 및 자궁각 용해물(b) 내 활성 PAI-1의 수준. 6시간 및 7일 시점에서, 활성 PAI-1 수준은 이소형 대조군 항체 처리 동물에 비해 A44V11 항체로 처리된 동물에서 복강 내 유체(IPF) 및 자궁각(UH) 용해물 모두에서 낮았고, 72시간 시점에서 차이가 관찰되지 않았다(독립표본 T-검정(Student T-test)에 의해 계산된 * p<0.001).
도 37은 동종 재조합 6-His 태그 Fab A44의 정제의 다른 예시를 도시한 것이다.
도 38은 인간 wt PAI-1 단백질과 복합된 동종 재조합 6-His 태그 Fab A44의 정제를 도시한 것이다.
도 39(a)는 Fab A44/PAI-1 복합체의 복합체 결정화를 도시한 것이고, 도 39(b)는 최적 결정을 도시한 것이다.
도 40은 Fab A44/PAI-1 복합체의 막대형 단일 결정을 도시한 것이다.
도 41은 인간 PAI-1의 활성 형태 및 시노 PAI-1의 잠복성 형태에 대한 Fab A44 인식을 도시한 것이다.
도 42는 (a) 활성 인간 PAI-1, 및 (b) 잠복성 시노 PAI-1 내 Fab A44에 의해 인식되는 PAI-1 에피토프를 도시한 것이다.
도 43은 Fab A44/PAI-1 복합체의 중쇄 파라토프를 도시한 것이다.
도 44는 Fab A44/PAI-1 복합체의 경쇄 파라토프를 도시한 것이다.
도 45는 시노, 인간, 랫트, 및 마우스 PAI-1의 제안된 A44 결합 에피토프의 서열 정렬을 도시한 것이다. 서열은 서열 번호 1(PAI-1 인간), 서열 번호 162(PAI-1 시노), 서열 번호 163(PAI-1 마우스), 및 서열 번호 164(PAI-1 랫트)로부터 발췌된다.
도 46은 마우스 PAI-1 구조와 인간 PAI-1/A44V11 복합체의 구조를 비교한 것을 도시한다.
도 47은 인간 PAI-1/A44V11 복합체의 구조 및 PAI-1에 대한 비브로넥틴(vibronectin) 결합의 모델을 보여준다.
도 48은 시노-PAI-1(서열 번호 162)의 펩신의 펩티드 커버리지를 도시한 것이다; 150개의 중첩되는 펩신의 펩티드로부터 95.3% 서열 커버리지가 얻어진다.
도 49는 미결합(원 선), APGv2-결합(x-선) 및 44v11-결합(다이아몬드 선) 상태에서 시노-PAI-1 펩티드에 대한 대표적인 중수소 흡수도를 도시한 것이다. 잔기 범위/위치는 서열 번호 162로부터 유래한다. (a) 대부분의 펩신의 펩티드는 시노-PAI-1 단독과 mAb에 결합한 것 사이에 차이를 나타내지 않았다. (b), 잔기 44~64를 포함하는 펩티드는 양자의 mAb-결합 상태에서 유사한 교환으로부터의 보호를 보였다. (c), 잔기 295~322를 포함하는 펩티드는 mAb-결합 상태 양자에서 적은 중수소를 혼입시키지만, A44v11에 대한 보호의 정도는 크다.
도 50은 A44v11에 결합한 시노-PAI-1 및 시노-PAI-1 단독의 수소/중수소 교환(HDX) 비교를 도시한 것이다. (a), 위쪽에는 미결합 상태 그리고 아래쪽에는 결합 상태인 평균 상대 분획 교환의 나비형 도표. 선은 10초, 1분, 5분 및 240분 시점에서 획득한 데이터에 상응한다. (b)에서, 시노-PAI-1 단독 또는 A44v11에 결합한 시노-PAI-1 (a)에서의 위의 도표와 상이한 데이터의 도표(달톤으로 나타냄).
도 51은 시노-PAI-1 단독 및 APGv2에 결합한 시노-PAI-1의 HDX 비교를 도시한 것이다. (a), 위쪽에 미결합 상태 및 아래쪽에 결합 상태를 가지는 평균 상대 분획 교환의 나비형 도표. 선은 10초, 1분, 5분 및 240분 시점에서 획득한 데이터에 상응한다. (b)에서, 시노-PAI-1 단독 또는 APGv2에 결합된 시노-PAI-1에 대한 위의 패널(a)로부터의 상이한 데이터에 대한 도표.
도 52는 A44v11에 결합된 시노-PAI-1 및 APGv2에 결합된 시노-PAI-1의 HDX 비교를 도시화한 것이다. (a), 위쪽에는 APGv2 결합 상태 및 아래쪽에는 A44v11 결합 상태를 가지는 평균 상대 분획 교환의 나비형 도표. 선은 10초, 1분, 5분 및 240분 시점에서 획득한 데이터에 상응한다. (b), 위의 패널(a)로부터의 APGv2에 결합한 시노-PAI-1 또는 A44v11에 결합한 시노-PAI-1에 대한 상이한 데이터의 도표.
도 53은 HDX MS에 의해 결정된 시노-PAI-1:A44v11 에피토프를 도시한 것이다. A44v11 항체에 결합된 상태에서 교환으로부터의 보호를 나타내는 시노 PAI-1(서열 번호 162)의 잔기는 굵은 글씨로 나타나 있다. 결정화 연구로부터 결정된 시노-PAI-1:A44v11 에피토프의 잔기가 상자에 나타나 있다.
도 2는 실시예 2에 기술된 바와 같은 결합 ELISA에서 항체 역가에 대한 통상적인 표준 곡선을 도시한 것이다. 항체 31C9, 33B8 및 33H1는 양성 대조군이었고 IgG1은 음성 대조군이었다.
도 3은 실시예 4에 기술된 바와 같이 tPA와 PAI-1의 상호작용을 차단하는 선택 항체에 대한 기능적 ELISA에 대한 대표 곡선을 도시한 것이다. 항체 33H1은 양성 대조군이고, IgG1는 음성 대조군이고 A44는 양성 항체 클론으로 확인되었다.
도 4는 실시예 4에 기술된 발색 검정에서 상업적으로 이용가능한 항체(33B8 및 33H1) 및 A44에 의한 tPA의 인간 PAI-1 차단 활성의 중화를 도시한 것이다.
도 5는 상이한 융합으로부터 생성된 항체의 선택에 의한 tPA의 인간 PAI-1 차단 활성의 중화를 도시한 것이다(실시예 4 참조).
도 6은 발색 검정에서 인간 PAI-1 및 그의 오소로그(ortholog)가 인간 tPA 활성을 유사한 효능으로 차단한다는 것을 도시한 것이다.
도 7은 실시예 4에 기술된 발색 검정에서 A44 및 33B8(상업적으로 이용가능함) 항체에 의한 인간 tPA의 시노몰구스(시노) 및 마우스 PAI-1 차단 활성의 중화를 도시한 것이다.
도 8은 항체 33H8(PAI-1을 활성 형태로부터 잠복성 형태로 전환함), 33H1(PAI-1을 활성 형태로부터 기질 형태로 전환함) 및 A44가 PAI-1과 tPA의 상호작용을 차단하는 작용의 기전의 SDS-Page 분석을 도시한 것이다. 레인 1: 분자량 기준; 레인 2: PAI-1 단독; 레인 3: tPA 단독; 레인 4: tPA의 존재 하 PAI-1; 레인 5: 33B8 +PAI-1 +tPA; 레인 6: 33H1+PAI-1+tPA; 레인 7: A44+PAI-1+tPA; 레인 8: mAb는 이소형 대조 항체이다.
도 9는 항체 33H8(PAI-1을 활성 형태로부터 잠복성 형태로 전환함), 33H1(PAI-1을 활성 형태로부터 기질 형태로 전환함) 및 융합 C26, E16 및 E21로부터 개발된 항체의 PAI-1과 tPA의 상호작용을 차단하기 위한 작용의 기전의 SDS-Page 분석을 도시한 것이다. 레인 1: 분자량 기준; 레인 2: PAI-1 단독; 레인 3: tPA 단독; 레인 4: tPA의 존재 하 PAI-1; 레인 5: 33B8 +PAI-1 +tPA; 레인 6: 33H1+PAI-1+tPA; 레인 7: C26+PAI-1+tPA; 레인 8: E16+PAI-1+tPA; 레인 9: E21+PAI-1+tPA; 레인 10: mAb는 이소형 대조 항체이다.
도 10은 항체 33H8(PAI-1을 활성 형태로부터 잠복성 형태로 전환함), 33H1(PAI-1을 활성 형태로부터 기질 형태로 전환함) 및 융합 A39, B109 및 C45로부터 개발된 항체의 PAI-1과 tPA의 상호작용을 차단하기 위한 작용의 기전의 SDS-Page 분석을 도시한 것이다. 레인 1: 분자량 기준; 레인 2: PAI-1 단독; 레인 3: tPA 단독; 레인 4: tPA의 존재 하 PAI-1; 레인 5: 33B8 +PAI-1 +tPA; 레인 6: 33H1+PAI-1+tPA; 레인 7: A39+PAI-1+tPA; 레인 8: B109+PAI-1+tPA; 레인 9: C45+PAI-1+tPA; 레인 10: mAb는 이소형 대조 항체이다.
도 11은 다음의 뮤라인 항체의 경쇄의 정렬을 도시한 것이다: A105(서열 번호 3), A39(서열 번호 5), A44(서열 번호 7), A71(서열 번호 9), A75(서열 번호 81), B109(서열 번호 11), B28(서열 번호 13), C45(서열 번호 15), E16(서열 번호 17), 및 E21(서열 번호 19). CDR은 굵은 글씨로 강조되어 있다.
도 12는 다음의 뮤라인 항체의 중쇄의 정렬을 도시한 것이다: A105(서열 번호 2), A39(서열 번호 4), A44(서열 번호 6), A71(서열 번호 8), A75(서열 번호 80), B109(서열 번호 10), B28(서열 번호 12), C45(서열 번호 14), E16(서열 번호 16), 및 E21(서열 번호 18). IMGT에 의해 정의된 CDR은 굵은 글씨로 강조되어 있다.
도 13은 뮤라인 A44 경쇄(서열 번호 7)와 vk1(서열 번호 101) 및 v람다3(서열 번호 102)의 정렬을 도시한 것이다.
도 14는 뮤라인 A44 중쇄(서열 번호 6)와 vh2(서열 번호 103) 및 vh4(서열 번호 104)의 정렬을 도시한 것이다.
도 15는 클론 A44 인간화 VL과 정렬된 모든 구조체(construct)를 도시한 것이다. 모든 정렬된 서열(서열 번호: 91~98)은 아래의 표 25에 추가로 기술되어 있다. 검은색 상자는 CDR 도메인을 나타낸다. 강조된 잔기는 정렬 내 바로 위의 잔기와 서열이 상이하다. 잔기 번호는 IMGT에 기술된 바와 같다.
도 16은 클론 A44 인간화 VH와 정렬된 모든 구조체를 도시한 것이다. 모든 정렬된 서열(서열 번호: 82~90)은 표 25에 추가로 기술되어 있다. 검은색 상자는 CDR 도메인을 나타낸다. 강조된 잔기는 정렬 내 바로 위의 잔기와 서열이 상이하다. 잔기 번호는 IMGT에 기술된 바와 같다.
도 17은 PAI-1 활성의 저해 백분율이 mAb 농도의 함수로서 도표화되었고, IC50은 Biostat 스피드 소프트웨어를 사용한 Imax로 결정되었다는 것을 도시한다.
도 18은 동종 재조합 6-His 태그 Fab A44의 정제를 도시한 것이다.
도 19는 고정화 APG 항체에 결합된 인간 PAI-1 글리코실화의 단일 운동 분석을 이용한 비아코어 2000으로의 SPR 분석을 도시한 것이다. 단일 주기 운동으로부터의 센서그램은 회색으로 나타낸다. 적합 모델(fit model)은 검은색으로 나타낸다.
도 20은 ELISA에 의한 UK-PAI-1 복합체 형성 검출에 의해 결정된 APG, APGv2, 및 APGv4 항체에 의한 인간 혈장 PAI-1 중화를 도시한 것이다. PAI-1 활성의 저해 백분율은 APG, APGv2, 또는 APGv4 항체의 농도의 함수로서 도표화되었다.
도 21은 시간(분)의 함수로서 340 nm에서의 흡광도 판독에 의한 혼탁 운동 측정으로 검출된 tPA 1 nM 및 PAI-1 3 nM의 존재 하에 A44V11(1, 3 또는 10 nM)에 의한 인간 혈장 응괴 용해의 회복을 도시한 것이다.
도 22는 시간(분)의 함수로서 340 nm에서의 흡광도로 검출된 tPA 1 nM 및 PAI-1 3 nM의 존재 하에 인간 IgG1 음성 대조군(1, 3 또는 10 nM)에 의해서는 인간 혈장 응괴 용해의 회복이 부재함을 도시한 것이다.
도 23은 A44V11 또는 다양한 농도의 인간 IgG1 이소형 음성 대조군에 의한 인간 혈장 응괴 용해의 회복을 도시한 것이다.
도 24는 시간(분)의 함수로 340 nm의 흡광도에서 검출된 tPA 1 nM 및 PAI-1 3 nM의 존재 하에 3 nM에서의 APG, APGV2 또는 APGV4에 의한 인간 혈장 응괴 용해의 회복을 도시한 것이다.
도 25는 다양한 농도의 APG 변이체 2 및 4에 의한 인간 혈장 응괴 용해의 회복을 도시한 것이다.
도 26은 TGFβ 5 ng/ml 및 50 nM의 IgG 이소형 대조군 mAb 또는 A44V11에 의한 처리 48H 후에서의 인간 LL29 근섬유아세포 상층액에 대한 면역블롯 항-PAI-1을 도시한 것이다.
도 27은 PBS(대조군), 플라스미노겐(Pg), A44v11 및 플라스미노겐(A+Pg) 또는 음성 인간 IgG 및 플라스미노겐(Neg+Pg)으로의 48시간 세포 처리 후 인간 일차 폐 섬유아세포 내 전체적인 MMP 활성을 도시한 것이다.
도 28은 4일에 복강 내 투여에 의한 A44 또는 IgG1(10 mg/kg) 또는 PBS로의 처리 후 7일 및 9일에서 블레오마이신 처리된 마우스로부터의 기관지 폐포 세척액(broncho-alveolar lavage fluid, BALF)(a) 및 폐 용해물(b) 내 인간 활성 PAI-1 수준을 도시한 것이다. 활성 PAI-1은 ELISA(# HPAIKT Molecular Innovation)에 의해 결정된다. 저해의 백분율은 A44 블레오와 IgG 블레오 사이의 차를 IgG 블레오 및 미처리(PBS) 마우스 군과의 차로 나눠서 계산하였다.
도 29는 ELISA(Asserachrom D-Di, Diagnostica Stago)에 의해 결정된 4일에복강 내 투여에 의한 A44 또는 IgG1(10 mg/kg) 또는 PBS로의 처리 후 7일 및 9일에서의 블레오마이신 처리된 마우스로부터의 BALF 내 마우스 D-이량체 수준을 도시한 것이다. A44에 의해 유도된 D-이량체 내 배율 증가는 IgG에 비교하여 나타낸다.
도 30은 4일부터 20일까지 3일마다 PBS(비히클), IgG1 또는 A44 10mg/kg 복강 내 투여가 이어지는 염수 또는 블레오마이신 처리 21일 후 유전자이식 인간화 마우스로부터의 우측 폐 중량을 도시한 것이다.
도 31은 4일부터 20일까지 3일마다 PBS(비히클), IgG1 또는 A44 10mg/kg 복강 내 투여가 이어지는 염수 또는 블레오마이신 처리 21일 후 유전자이식 인간화 마우스 내 하이드록시프롤린 폐 함량을 도시한 것이다.
도 32는 LPS 챌린지(100 ug/kg iv) 24시간 전에 A44V11(a) mAb(n=5) 또는 IgG1 이소형 대조군(b)(n=4)(5 mg/kg ip)으로 처리된 원숭이로부터의 혈장 내 활성 PAI-1 수준을 도시한 것이다. 혈액 시료를 지시된 시점에서 수확하였고, 활성 PAI-1 수준을 혈장에서 ELISA(Molecular Innovation으로부터의 # HPAIKT)를 사용하여 결정하였다.
도 33은 LPS 챌린지(100 ug/kg iv) 24시간 전에 A44V11(a) mAb(n=5) 또는 IgG1 이소형 대조군(b)(n=4)(5 mg/kg ip)으로 처리된 원숭이로부터의 간 생검물 내 활성 PAI-1 수준을 도시한 것이다. 간 생검물을 지시된 시점에 마취된 원숭이에서 수확하였고, 용해물 내 활성 PAI-1 수준을 ELISA(Molecular Innovation # HPAIKT)를 사용하여 결정하였다.
도 34는 LPS 챌린지(100 ug/kg iv) 24시간 전에 A44V11(a) mAb(n=5) 또는 IgG1 이소형 대조군(b)(n=4)(5 mg/kg ip)으로 처리된 원숭이로부터의 혈장 내 D-이량체 수준을 도시한 것이다. 혈액 시료를 지시된 시점에서 수확하였고, D-이량체 수준을 혈장에서 ELISA를 사용하여 결정하였다.
도 35는 LPS 챌린지(100 ug/kg iv) 24시간 전에 A44V11(a) mAb(n=5) 또는 IgG1 이소형 대조군(b)(n=4)(5 mg/kg ip)으로 처리된 원숭이로부터의 혈장 내 플라스민-α2 항플라스민(PAP) 복합체 수준을 도시한 것이다. 혈액 시료를 지시된 시점에서 수확하였고, PAP 수준을 혈장에서 ELISA(Diagnostica Stago로부터의 # Asserachrom PAP)를 사용하여 결정하였다.
도 36은 복강 내 유체(intraperitoneal fluid, IPF) 및 자궁각 용해물 내 활성 PAI-1의 수준을 도시한 것이다. 복강 내 유체(intraperitoneal fluid, IPF)(a) 및 자궁각 용해물(b) 내 활성 PAI-1의 수준. 6시간 및 7일 시점에서, 활성 PAI-1 수준은 이소형 대조군 항체 처리 동물에 비해 A44V11 항체로 처리된 동물에서 복강 내 유체(IPF) 및 자궁각(UH) 용해물 모두에서 낮았고, 72시간 시점에서 차이가 관찰되지 않았다(독립표본 T-검정(Student T-test)에 의해 계산된 * p<0.001).
도 37은 동종 재조합 6-His 태그 Fab A44의 정제의 다른 예시를 도시한 것이다.
도 38은 인간 wt PAI-1 단백질과 복합된 동종 재조합 6-His 태그 Fab A44의 정제를 도시한 것이다.
도 39(a)는 Fab A44/PAI-1 복합체의 복합체 결정화를 도시한 것이고, 도 39(b)는 최적 결정을 도시한 것이다.
도 40은 Fab A44/PAI-1 복합체의 막대형 단일 결정을 도시한 것이다.
도 41은 인간 PAI-1의 활성 형태 및 시노 PAI-1의 잠복성 형태에 대한 Fab A44 인식을 도시한 것이다.
도 42는 (a) 활성 인간 PAI-1, 및 (b) 잠복성 시노 PAI-1 내 Fab A44에 의해 인식되는 PAI-1 에피토프를 도시한 것이다.
도 43은 Fab A44/PAI-1 복합체의 중쇄 파라토프를 도시한 것이다.
도 44는 Fab A44/PAI-1 복합체의 경쇄 파라토프를 도시한 것이다.
도 45는 시노, 인간, 랫트, 및 마우스 PAI-1의 제안된 A44 결합 에피토프의 서열 정렬을 도시한 것이다. 서열은 서열 번호 1(PAI-1 인간), 서열 번호 162(PAI-1 시노), 서열 번호 163(PAI-1 마우스), 및 서열 번호 164(PAI-1 랫트)로부터 발췌된다.
도 46은 마우스 PAI-1 구조와 인간 PAI-1/A44V11 복합체의 구조를 비교한 것을 도시한다.
도 47은 인간 PAI-1/A44V11 복합체의 구조 및 PAI-1에 대한 비브로넥틴(vibronectin) 결합의 모델을 보여준다.
도 48은 시노-PAI-1(서열 번호 162)의 펩신의 펩티드 커버리지를 도시한 것이다; 150개의 중첩되는 펩신의 펩티드로부터 95.3% 서열 커버리지가 얻어진다.
도 49는 미결합(원 선), APGv2-결합(x-선) 및 44v11-결합(다이아몬드 선) 상태에서 시노-PAI-1 펩티드에 대한 대표적인 중수소 흡수도를 도시한 것이다. 잔기 범위/위치는 서열 번호 162로부터 유래한다. (a) 대부분의 펩신의 펩티드는 시노-PAI-1 단독과 mAb에 결합한 것 사이에 차이를 나타내지 않았다. (b), 잔기 44~64를 포함하는 펩티드는 양자의 mAb-결합 상태에서 유사한 교환으로부터의 보호를 보였다. (c), 잔기 295~322를 포함하는 펩티드는 mAb-결합 상태 양자에서 적은 중수소를 혼입시키지만, A44v11에 대한 보호의 정도는 크다.
도 50은 A44v11에 결합한 시노-PAI-1 및 시노-PAI-1 단독의 수소/중수소 교환(HDX) 비교를 도시한 것이다. (a), 위쪽에는 미결합 상태 그리고 아래쪽에는 결합 상태인 평균 상대 분획 교환의 나비형 도표. 선은 10초, 1분, 5분 및 240분 시점에서 획득한 데이터에 상응한다. (b)에서, 시노-PAI-1 단독 또는 A44v11에 결합한 시노-PAI-1 (a)에서의 위의 도표와 상이한 데이터의 도표(달톤으로 나타냄).
도 51은 시노-PAI-1 단독 및 APGv2에 결합한 시노-PAI-1의 HDX 비교를 도시한 것이다. (a), 위쪽에 미결합 상태 및 아래쪽에 결합 상태를 가지는 평균 상대 분획 교환의 나비형 도표. 선은 10초, 1분, 5분 및 240분 시점에서 획득한 데이터에 상응한다. (b)에서, 시노-PAI-1 단독 또는 APGv2에 결합된 시노-PAI-1에 대한 위의 패널(a)로부터의 상이한 데이터에 대한 도표.
도 52는 A44v11에 결합된 시노-PAI-1 및 APGv2에 결합된 시노-PAI-1의 HDX 비교를 도시화한 것이다. (a), 위쪽에는 APGv2 결합 상태 및 아래쪽에는 A44v11 결합 상태를 가지는 평균 상대 분획 교환의 나비형 도표. 선은 10초, 1분, 5분 및 240분 시점에서 획득한 데이터에 상응한다. (b), 위의 패널(a)로부터의 APGv2에 결합한 시노-PAI-1 또는 A44v11에 결합한 시노-PAI-1에 대한 상이한 데이터의 도표.
도 53은 HDX MS에 의해 결정된 시노-PAI-1:A44v11 에피토프를 도시한 것이다. A44v11 항체에 결합된 상태에서 교환으로부터의 보호를 나타내는 시노 PAI-1(서열 번호 162)의 잔기는 굵은 글씨로 나타나 있다. 결정화 연구로부터 결정된 시노-PAI-1:A44v11 에피토프의 잔기가 상자에 나타나 있다.
본 발명은 인간 PAI-1에 특이적으로 결합하고 PAI-1의 생물학적 기능을 조절하는 항체 및 이의 단편을 제공한다. 이러한 항체는 PAI-1 관련 질환 또는 장애(예를 들어, 섬유증)를 치료하는 데에 특히 유용하다. 본 발명은 또한 약학적 조성물뿐 아니라 PAI-1 항체를 암호화하는 핵산, 이러한 항체, 또는 이의 단편을 제조하기 위한 재조합 발현 벡터 및 숙주 세포도 제공한다. 시험관 내 또는 생체내에서 PAI-1을 검출하거나 PAI-1 활성을 조절하기 위해 본원에 개시된 항체를 사용하는 방법도 또한 본 발명에 포함된다.
I. 정의
본 발명을 보다 쉽게 이해할 수 있도록 하기 위해, 우선 일정한 용어들이 정의된다.
본원에서 사용된, "인간 PAI-1"이라는 용어는 하기에 열거된 아미노산 서열로 이루어지거나 이를 포함하는 펩티드를 지칭한다:
VHHPPSYVAHLASDFGVRVFQQVAQASKDRNVVFSPYGVASVLAMLQLTTGGETQQQIQAAMGFKIDDKGMAPALRHLYKELMGPWNKDEISTTDAIFVQRDLKLVQGFMPHFFRLFRSTVKQVDFSEVERARFIINDWVKTHTKGMISNLLGKGAVDQLTRLVLVNALYFNGQWKTPFPDSSTHRRLFHKSDGSTVSVPMMAQTNKFNYTEFTTPDGHYYDILELPYHGDTLSMFIAAPYEKEVPLSALTNILSAQLISHWKGNMTRLPRLLVLPKFSLETEVDLRKPLENLGMTDMFRQFQADFTSLSDQEPLHVAQALQKVKIEVNESGTVASSSTAVIVSARMAPEEIIMDRPFLFVVRHNPTGTVLFMGQVMEP(서열 번호 1), 또는 이의 단편.
본원에서 사용된, "항체"라는 용어는 이황화 결합에 의해 상호연결된 2개의 중(H)쇄 및 2개의 경(L)쇄인 4개의 폴리펩티드 사슬뿐 아니라 이의 다량체(예를 들어, IgM)를 포함하는 면역글로불린 분자를 지칭한다. 각각의 중쇄는 중쇄 가변 영역(VH 또는 VH로 축약됨) 및 중쇄 불변 영역(CH 또는 CH)을 포함한다. 중쇄 불변 영역은 CH1, CH2 및 CH3인 3개의 도메인을 포함한다. 각각의 경쇄는 경쇄 가변 영역(VL 또는 VL로 축약됨) 및 경쇄 불변 영역(CL 또는 CL)을 포함한다. 경쇄 불변 영역은 하나의 도메인(CL1)을 포함한다. VH 및 VL 영역은 구조형성 영역(FR)이라고 불리는 보다 보존된 영역 사이에 산재된 상보성 결정 영역(CDR)이라고 불리는 과가변성의 영역으로 더 나누어질 수 있다. 각각의 VH 및 VL은 아미노 말단에서부터 카복시 말단으로 다음의 순서로 배열된 3개의 CDR 및 4개의 FR로 구성된다: FR1, CDR1, FR2, CDR2, FR3, CDR3, FR4.
본원에서 사용된, 항체의 "항원 결합 단편"이라는 용어는 항원에 특이적으로 결합하여 복합체를 형성하는 임의의 자연적으로 발생하는, 효소적으로 얻어지는, 합성 또는 유전적으로 조작된 폴리펩티드 또는 당단백질을 포함한다. 항체의 항원 결합 단편은, 예를 들어, 항체 가변 및 선택적으로 불변 도메인을 암호화하는 DNA의 발현 및 조작을 수반하는 재조합 유전자 조작 기술 또는 단백질 가수분해성 분해와 같은 임의의 적합한 표준 기술을 사용하여 전체 항체 분자로부터 유래될 수 있다. 항원 결합 부위의 비제한적인 예는 (i) Fab 단편; (ii) F(ab')2 단편; (iii) Fd 단편; (iv) Fv 단편; (v) 단쇄 Fv(scFv) 분자; (vi) dAb 단편; 및 (vii) 항체의 과가변 영역(예를 들어, 단리된 상보성 결정 영역(CDR))을 모사하는 아미노산 잔기로 이루어진 최소 인식 단위를 포함한다. 다른 조작된 분자, 예를 들어, 디아바디(diabody), 트리아바디(triabody), 테트라바디(tetrabody) 및 미니바디(minibody)도 또한 "항원 결합 단편" 발현 내에 포함된다.
본원에서 사용된, "CDR" 또는 "상보성 결정 영역"이라는 용어는 중쇄 및 경쇄 폴리펩티드의 가변 영역 내에서 발견되는 비연속적인 항원 결합 자리를 의미한다. 이러한 특정 영역은 문헌(Kabat et al., J. Biol . Chem. 252, 6609-6616(1977) 및 Kabat et al., Sequences of protein of immunological interest.(1991), 및 Chothia et al., J. Mol . Biol. 196:901-917(1987) 및 MacCallum et al., J. Mol . Biol. 262:732-745(1996))에 기술되어 있는데, 여기서 이러한 정의는 서로에 대해 비교할 때 아미노산 잔기의 하위 세트 또는 중첩을 포함한다. 카바트(Kabat)의 정의는 서열 가변성을 기반으로 한다. 모든 종의 모든 IG 및 TR V-영역에 대한 IMGT 고유 번호는 가변 영역의 구조의 높은 보존성에 의존한다(Lefranc, Mp et al., Dev comp. Immunol. 27:55-77, 2003). 5,000개를 초과하는 서열의 정렬 후 설정된 IMGT 번호가 고려되고 구조형성 및 CDR의 정의와 조합된다. 클로티아(Clothia) 정의는 구조적 루프 영역의 위치를 기반으로 한다. 접촉 정의(MacCallum et al.)는 복합체 결정 구조 및 항체-항원 상호작용의 분석을 기반으로 한다. 위에 인용된 참조문헌 각각에 의해 정의된 바와 같은 CDR을 포함하는 아미노산 잔기가 비교를 위해 기술되어 있다. 본원에 개시된 일 구현예에서, "CDR"이라는 용어는 카바트 정의에 의해 정의된 바와 같은 CDR이다. 본원에 개시된 다른 구현예에서, CDR은 IMGT에 의해 정의된 바와 같은 CDR이다.
본원에서 사용된 "구조형성(FR) 아미노산 잔기"라는 용어는 Ig 사슬의 구조형성 영역 내의 이러한 아미노산을 지칭한다. 본원에서 사용된 "구조형성 영역" 또는 "FR 영역"이라는 용어는, 가변 영역의 일부이지만, (예를 들어, CDR의 접촉 정의를 사용한) CDR의 일부는 아닌 아미노산 잔기를 포함한다. 따라서, 가변 영역 구조형성은 길이가 약 100~120 아미노산 사이이지만, CDR의 외부의 아미노산만을 포함한다.
본 발명은 또한 본원에 개시된 항체의 CDR 아미노산 서열 내 "보존적 아미노산 치환", 즉, 항원, 즉, PAI-1에 대한 항체의 결합을 방해하지 않는 아미노산 서열 변형을 포함한다. 보존적 치환은 이러한 위치에서 아미노산 잔기의 전하 또는 극성에 대한 영향이 거의 없거나 전혀 없는 원래의 아미노산 잔기의 비자연적 잔기로의 치환이다. 예를 들어, 보존적 치환은 폴리펩티드 내 비극성 잔기의 임의의 다른 비극성 잔기로의 교체로 인한 보존적 치환이다. 나아가, 폴리펩티드 내 임의의 원래의 잔기는 또한 "알라닌 스캐닝 돌연변이유발"(Cunningham et al., Science 244:1081-85(1989))에 대해 이전에 기술된 바와 같이, 알리닌으로 치환될 수 있다. 보존적 아미노산 치환은 하나의 클래스 내 아미노산의 동일한 클래스의 아미노산으로의 치환을 포함하되, 여기서 클래스는, 예를 들어, 표준 데이호프 빈도 교환 매트릭스(Dayhoff frequency exchange matrix) 또는 BLOSUM 매트릭스에 의해 결정된 자연계에서 발견되는 상동 단백질 내 높은 치환 빈도 및 공통적인 물리화학적 아미노산 측쇄 성질에 의해 정의된다. 6개의 일반적인 클래스의 아미노산 측쇄가 목록화 되어 있으며, 클래스 I(Cys); 클래스 II(Ser, Thr, Pro, Ala, Gly); 클래스 III(Asn, Asp, Gln, Glu); 클래스 IV(His, Arg, Lys); 클래스 V(Ile, Leu, Val, Met); 및 클래스 VI(Phe, Tyr, Trp)를 포함한다. 예를 들어, Asp의, 다른 클래스 III 잔기, 예를 들어, Asn, Gln, 또는 Glu으로의 치환은 보존적 치환이다. 따라서, PAI-1 항체 내에서 예측된 비필수 아미노산 잔기는 동일한 클래스의 다른 아미노산 잔기로 교체된다. 항원 결합을 제거하지 않는 아미노산 보존적 치환을 확인하는 방법은 당해 분야에 잘 알려져 있다(예를 들어, Brummell et al., Biochem. 32:1180, 1993; Kobayashi et al. protein Eng. 12:879, 1999; 및 Burks et al. Proc . Natl . Acad. Sci . USA 94:412, 1997 참조). 보존적 아미노산 치환에 대한 일반적인 규칙은 아래의 표 1에 기술되어 있다.
보존적 아미노산 치환은 또한 비자연적으로 발생하는 아미노산 잔기(통상적으로 생물학적 시스템 내에서의 합성보다는 화학적 펩티드 합성에 의해 삽입됨)도 포함한다. 이는 펩티도미메틱, 및 기타 아미노산 모이어티의 역전 또는 반전 형태를 포함한다.
아미노산 서열에 대한 보존적 변형(및 암호화 핵산에 대한 상응하는 변형)은 자연적으로 발생하는 PAI-1 항체의 것과 유사한 기능적 및 화학적 특성을 가지는 PAI-1 항체를 생성할 것으로 예상된다. 반대로, PAI-1 항체의 기능적 또는 화학적 특성에서의 실질적인 변형은 (a) 치환된 면적 내 분자 골격의 구조, 예를 들어, 시트 또는 나선형 형태, (b) 표적 자리에서의 분자의 전하 또는 소수성, 또는 (c) 측쇄의 벌크를 유지하는 효과가 유의하게 달라지는 치환을 선택함으로써 달성할 수 있다. 자연적으로 발생하는 잔기는 통상적인 측쇄 성질에 따라 군으로 분류될 수 있다:
1) 소수성: 노르류신, Met, Ala, Val, Leu, Ile;
2) 중성 친수성: Cys, Ser, Thr;
3) 산성: Asp, Glu;
4) 염기성: Asn, Gln, His, Lys, Arg;
5) 사슬 배향에 영향을 주는 잔기: Gly, Pro; 및
6) 방향족: Trp, Tyr, Phe.
비보존적 치환은 이러한 클래스 중 하나의 구성원의 다른 클래스로부터의 구성원으로의 교환에 관련될 수 있다. 이러한 치환된 잔기가 비인간 PAI-1 항체와 상동성인 인간 PAI-1 항체의 영역 내 또는 분자의 비상동성 영역 내에 도입될 수 있다.
일정한 양태에서, 중쇄 또는 경쇄 가변 영역은 본원에 개시된 임의의 가변 영역 서열에 99%, 98%, 97%, 96%, 95%, 94%, 93%, 92%, 91%, 또는 90% 동일하다.
본원에서 사용된, "특이적으로 결합한다"라는 용어는 1 x 10-6 M, 1 x 10-7 M, 1 x 10-8 M, 1 x 10-9 M, 1 x 10-10 M, 1 x 10-11 M, 1 x 10- 12이하보다 낮은 Kd로 항원에 결합하는 항체 또는 이의 항원 결합 단편의 능력을 지칭한다. 이러한 용어는 또한 비특이적 항원에 대한 친화도보다 적어도 2배 더 높은 친화도로 항원에 결합하는 항체 또는 이의 항원 결합 단편의 능력에 대한 지칭을 포함한다.
본 개시는 또한 당해 분야에 알려진 경쟁적 결합을 결정하기 위한 임의의 방법, 예를 들어, 본원에 기술된 면역검정법에 의해 결정된 본원에 개시된 에피토프에 대한 항체의 결합을 경쟁적으로 저해하는 항체를 제공한다. 일정한 구현예에서, 항체는 에피토프에 대한 결합을 적어도 95%, 적어도 90%, 적어도 85%, 적어도 80%, 적어도 75%, 적어도 70%, 적어도 60%, 또는 적어도 50%만큼 경쟁적으로 저해한다.
본원에서 사용된, "항원"이라는 용어는 항체 또는 이의 항원 결합 단편에 의해 인식되는 결합 자리 또는 에피토프를 지칭한다.
본원에서 사용된, "벡터”라는 용어는 핵산 분자가 연결되는 다른 핵산을 이동시킬 수 있는 핵산 분자를 지칭하는 것으로 의도된다. 벡터의 한 가지 유형은 "플라스미드”이며, 이것은 추가적인 DNA 세그먼트가 라이게이션될 수 있는 환형 이중 가닥 DNA 루프를 지칭한다. 다른 유형의 벡터는 바이러스 벡터로, 바이러스 게놈 내에 추가적인 DNA 세그먼트가 라이게이션될 수 있다. 일정 벡터(예컨대, 박테리아 복제 원점을 가지는 박테리아 벡터 및 포유동물 에피솜 벡터)는 그들이 도입되는 숙주 세포 내에서 자발적으로 복제할 수 있다. 다른 벡터(예컨대, 포유동물 비에피솜 벡터)들은 숙주 세포로의 도입 시 숙주 세포의 게놈 내로 삽입될 수 있어서 숙주 게놈과 함께 복제된다. 또한, 일정 벡터들은 그들이 작동가능하게 연결되는 유전자의 발현을 유도할 수 있다. 이러한 벡터들은 본원에서 "재조합 발현 벡터”(또는 간단하게, "발현 벡터”)로 지칭된다. 일반적으로, 재조합 DNA 기술에 활용되는 발현 벡터들은 대개 플라스미드의 형태이다. "플라스미드” 및 "벡터”라는 용어는 상호호환 가능하게 사용될 수 있다. 그러나, 본 발명은, 동등한 기능을 수행하는 바이러스 벡터(예컨대, 복제 결함 레트로바이러스, 아데노바이러스 및 아데노 관련 바이러스)와 같은 다른 형태의 발현 벡터들도 포함하는 것으로 의도된다.
다수의 발현 벡터 시스템이 본 발명의 목적을 위해 채용될 수 있다. 예를 들어, 하나의 클래스의 벡터는 소 파필로마 바이러스, 폴리오마 바이러스, 아데노바이러스, 백시니아 바이러스, 배큘로바이러스, 레트로바이러스(RSV, MMTV 또는 MOMLV) 또는 SV40 바이러스와 같은 동물 바이러스로부터 유래된 DNA 구성요소를 활용할 수 있다. 그 외는 내부 리보솜 결합 자리를 가지는 폴리시스트론 시스템의 이용에 관련된다. 추가적으로, DNA가 그들의 염색체 내로 삽입된 세포를 형질감염된 숙주 세포의 선택을 가능하게 하는 하나 이상의 마커를 도입함으로써 선택할 수 있다. 이러한 마커는 영양요구성 숙주에 독립영양을 제공하거나, 살생물제(예를 들어, 항생제) 내성, 또는 구리와 같은 중금속에 대한 내성을 제공할 수 있다. 선택적인 마커 유전자는 발현될 DNA 서열에 직접 연결되거나 공동형질전환에 의해 동일한 세포 내로 도입될 수 있다. 추가적인 구성요소들도 또한 mRNA의 최적 합성을 위해 필요할 수 있다. 이러한 구성요소들은 신호 서열, 스플라이싱 신호뿐 아니라 전사 프로모터, 인핸서, 및 종결 신호를 포함할 수 있다. 특정한 구현예에서 클로닝된 가변 영역 유전자는 위에서 논의된 바와 같이 중쇄 및 경쇄 불변 영역 유전자(예를 들어, 인간) 합성체를 따라 발현 벡터 내에 삽입된다.
보다 일반적으로, 일단 항체, 또는 이의 단편을 암호화하는 벡터 또는 DNA 서열이 제조되면, 발현 벡터는 적절한 숙주 세포 내로 도입될 수 있다. 즉, 숙주 세포가 형질전환될 수 있다. 숙주 세포로의 플라스미드의 도입은 당업자에게 잘 알려진 다양한 기술에 의해 달성될 수 있다. 이러한 기술은, 이에 제한되지는 않으나, 형질감염(전기영동 및 전기천공을 포함함), 원형질 융합, 인산 칼슘 침전, 외피 DNA를 이용한 세포 융합, 미세주입, 및 완전한 바이러스로의 감염을 포함한다. 문헌(Ridgway, A. A. G. "Mammalian expression vectors" Chapter 24.2, pp. 470-472 vectors, Rodriguez and Denhardt, Eds.(Butterworths, Boston, Mass. 1988))을 참조한다. 본원에 개시된 하나의 구현예는 전기천공을 통한 플라스미드의 숙주로의 도입이다. 형질전환된 세포를 경쇄 및 중쇄의 생성에 적절한 조건 하에 성장시키고, 중쇄 또는 경쇄 단백질 합성에 대해 분석한다. 예시적인 검정 기술은 효소 결합 면역 흡착 검정법(ELISA), 방사면역검정법(RIA), 또는 형광 활성화 세포 분류 분석법(FACS), 면역조직화학법 등을 포함한다.
본원에서 사용된, "형질전환"이라는 용어는 유전자형을 바꾸고 이에 따라 수여자 세포 내 변화를 초래하는, 수여자 숙주 세포 내로의 DNA의 도입을 지칭하는 넓은 의미로 사용되어야 한다.
"숙주 세포"는 재조합 DNA 기술을 사용하여 제작되고 적어도 하나의 이종 유전자를 암호화하는 벡터로 형질전환된 세포를 지칭한다. 재조합 숙주로부터의 폴리펩티드의 단리를 위한 과정의 설명에서, "세포" 및 "세포 배양물"이라는 용어는, 명백하게 다르게 명시되지 않는 한, 항체의 공급원을 나타내기 위해 상호교환가능하게 사용된다. 다시 말해, "세포"로부터 폴리펩티드의 회복은 전체 세포를 스핀 다운하거나 배지 및 현탁된 세포를 함유하는 세포 배양물로부터 유래된 것을 의미할 수 있다.
이러한 용어는 특정한 대상 세포뿐 아니라 이러한 세포의 자손세포도 지칭하는 것으로 의도된다는 것을 이해해야 한다. 돌연변이 또는 환경적 영향으로 인해, 계대에서 일정한 변형이 발생할 수 있기 때문에, 이러한 자손세포는, 실제로, 모 세포와 동일하지 않을 수 있지만, 본원에 사용된 "숙주 세포”라는 용어의 범주 내에 여전히 포함된다.
본원에서 사용된 "치료하다”, "치료하는” 및 "치료”라는 용어는 본원에 기술된 치료적 또는 예방적 조치를 지칭한다. "치료”의 방법은 질환 또는 장애 또는 재발한 질환 또는 장애의 하나 이상의 증상을 개선하거나 중증도를 감소시키거나 지연시키거나, 치료하거나, 예방하거나, 이러한 치료의 부재 시 예상되는 생존률보다 대상체의 생존률을 연장시키기 위해, 본원에 개시된 항체 또는 항원 결합 단편을 대상체, 예를 들어 PAI-I 관련 질환 또는 장애(예컨대, 섬유조직 질환)을 가지거나 이러한 질환 또는 장애를 가지는 경향을 가지는 대상체에 투여하는 것을 채용한다.
본원에 사용된 "PAI-I 관련 질환 또는 장애”라는 용어는 변형된 PAI-I의 수준 또는 활성이 발견되는 질환 상태에 연관된 증상을 가지거나 가지지 않는 질환 상태를 포함한다. 예시적인 PAI-I 관련 질환 또는 장애는 다양한 유형의 섬유증을 포함한다.
본원에 사용된 "유효량”이라는 용어는, 본원에 기술된 바와 같이, 대상체에 투여될 때, PAI-I 관련 질환 또는 장애의 치료, 예후, 또는 진단을 시행하기에 충분한 PAI-I에 결합하는 항체 또는 이의 항원 결합 단편의 양을 지칭한다. 치료적 유효량은 치료될 병태 및 대상체, 대상체의 체중 및 연령, 병태의 중증도, 투여의 방식 등에 따라 달라질 것이고, 당업자에 의해 용이하게 결정될 수 있다. 투여량은, 예를 들어, 약 1 ng 내지 약 10,000 mg, 약 1 ug 내지 약 5,000 mg, 약 1 mg 내지 약 1,000 mg, 약 10 mg 내지 약 100 mg의 범위의 본원에 개시된 항체 또는 이의 항원 결합 단편일 수 있다. 투여 계획은 최적 치료 반응을 제공하기 위해 조정될 수 있다. 또한, 유효량은 항체 또는 이의 항원 결합 단편의 임의의 독성 또는 해로운 효과(즉, 부작용)가 최소화되거나 이로운 효과가 더 큰 양이다.
본원에서 사용된 "대상체”또는 "포유동물"이라는 용어는 임의의 인간 또는 인간이 아닌 동물을 포함한다.
본원에서 사용된 "에피토프”라는 용어는 파라토프(paratope)라고 알려진 항체 분자의 가변 영역 내 특이적인 항원 결합 자리와 상호작용하는 항원성 결정기를 지칭한다. 단일 항원이 하나 이상의 에피토프를 가질 수 있다. 따라서, 상이한 항체들은 항원 상의 상이한 영역에 결합할 수 있고, 상이한 생물학적 효과를 가질 수 있다. 에피토프는 구조적 또는 선형일 수 있다. 구조적 에피토프는 선형 폴리펩티드 사슬의 상이한 세그먼트로부터 공간적으로 나란히 놓인 아미노산에 의해 생성된다. 선형 에피토프는 폴리펩티드 사슬 내 인접한 아미노산 잔기에 의해 생성되는 것이다.
여기서, 본 명세서 및 첨부된 청구범위에서 사용된 "하나의(a, an, the)"와 같은 단수 형태는 문맥에서 명확하게 다르게 지시되지 않는 한 복수 참조를 포함한다는 것을 주지한다.
II. 항-
PAI
-
1항체
일 양태에서 본 발명은 인간 PAI-1에 특이적으로 결합하는 항체, 또는 이의 항원 결합 단편을 제공한다. 본원에 개시된 항체의 예시적인 VH, VL 및 CDR 아미노산 서열 및 뉴클레오티드 서열이 표 2에 기술되어 있다. 표 2에 나타낸 CDR 영역은 IMGT에 의해 정의된다.
다른 구현예에서, 본 발명은, 각각, 서열 번호 6 및 7에 기재된 VH 및 VL 영역 아미노산 서열을 포함하는 항체, 또는 이의 항원 결합 단편을 경쟁적으로 저해하거나 이와 동일한 에피토프에 결합하는 항-PAI-1 항체를 제공한다. 이러한 항체는, 예를 들어, 표면 플라스몬 공명(SPR)-기반 경쟁 검정을 포함하는, 일상적인 경쟁 결합 검정법을 사용하여 확인할 수 있다.
III. 변형된 항-
PAI
-1 항체
일정한 구현예에서, 본원에 개시된 항-PAI-1 항체는 하나 이상의 변형을 포함할 수 있다. 본원에 개시된 항-PAI-1 항체의 변형된 형태는 당해 분야에 알려진 임의의 기술을 사용하여 제조될 수 있다.
i) 면역원성 감소
일정 구현예에서, 본원에 개시된 PAI-1 항체 또는 이의 항원 결합 단편은 그들의 면역원성을 낮추기 위해 당해 분야에 알려진 기술을 사용하여 변형된다. 예를 들어, 항체 또는 이의 단편은 키메라화되거나, 인간화되거나, 탈면역화될 수 있다.
일 구현예에서, 본원에 개시된 항체 또는 이의 항원 결합 단편은 키메라일수 있다. 키메라 항체는 항체의 상이한 부위들이 상이한 동물 종으로부터 유래한 항체, 예를 들면 뮤라인 단일클론 항체로부터 유래한 가변 영역 및 인간 면역글로불린 불변 영역을 가지는 항체이다. 키메라 항체 또는 이의 단편을 제조하기 위한 방법은 당해 분야에 공지되어 있다. 예컨대, 그 전체가 본원에 참조로서 포함된 문헌(예를 들어, Morrison, Science 229:1202 1985; Oi et al., BioTechniques 4:214 (1986); Gillies et al., J. Immunol . Methods 125:191 1989; 미국 특허 제5,807,715호; 제4,816,567호; 및 제4,816,397호)을 참조한다. "키메라 항체"의 생성을 위해 개발된 기술(Morrison et al., Proc . Natl . Acad . Sci. 81:851-855 1984; Neuberger et al., Nature 312:604-608 1984; Takeda et al., Nature 314:452-454 1985)이 상기 분자의 합성을 위해 채용될 수 있다. 예를 들어, 마우스 PAI-1 항체 분자의 결합 특이성을 암호화하는 유전 서열이 적절한 생물학적 활성의 인간 항체 분자로부터의 서열과 함께 융합될 수 있다. 본원에서 사용된 바와 같이, 키메라 항체는 상이한 부위들이 상이한 동물종으로부터 유래된 분자, 예를 들면 뮤라인 단일클론 항체로부터 유래된 가변 영역 및 인간 면역글로불린 불변 영역을 가지는 분자, 예컨대, 인간화 항체이다.
다른 구현예에서, 본원에 개시된 항체 또는 이의 항원 결합 단편은 인간화된다. 인간화 항체는 비인간 항체로부터의 하나 이상의 상보성 결정 영역(CDR) 및 인간 항체 분자로부터의 구조형성 영역을 포함하는 결합 특이성을 가진다. 종종, 인간 구조형성 영역의 구조형성 잔기는 항원 결합을 변경 또는 개선하기 위해 CDR 공여자 항체로부터의 상응하는 잔기로 치환될 것이다. 이러한 구조형성부 치환은 당해분야에 잘 공지된 방법에 의해, 예컨대 항원 결합에 중요한 구조형성 잔기를 식별하기 위한 CDR과 구조형성 잔기의 상호작용의 모델링 및 특정 위치에서 특이한 구조형성 잔기를 식별하기 위한 서열비교에 의해 식별된다. 예컨대, 그 전체가 본원에 참조로서 포함된 Queen et al., 미국 특허 제5,585,089호; Riechmann et al., Nature 332:323 1988)을 참조한다. 항체는, 예를 들어 CDR 이식(EP 239,400; 국제 공개 WO 91/09967; 미국 특허 제5,225,539호; 제5,530,101호; 및 제5,585,089호), 베니어링(veneering) 또는 표면치환(resurfacing)(EP 592,106; EP 519,596; Padlan, Molecular Immunology 28:489-498, 1991; Studnicka et al., Protein Engineering 7:805, 1994; Roguska. et al., PNAS 91:969, 1994) 및 사슬 셔플링(미국 특허 제5,565,332호)을 포함하는 당해 분야에 공지된 다양한 기술들을 사용하여 인간화될 수 있다.
특정 구현예에서, 면역 인식 중간 및 면역 인식에서 항체의 분자적 유연성의 영향에 기반한 인간화 방법이 채용된다(그 전체가 본원에 참조로서 포함된 국제 공개 번호 WO2009/032661을 참조한다). 단백질 유연성은 단백질 분자의 분자 운동에 관련된다. 단백질 유연성은 전체 단백질, 단백질의 일부, 또는 단일 아미노산 잔기가 서로 현저하게 다른 구조들의 총체를 채택하는 능력이다. 단백질 유연성에 대한 정보는 단백질 X-선 결정 실험(예를 들어, Kundu et al., Biophys . J. 83:723, 2002을 참조), 핵자기공명 실험(예를 들어, Freedberg et al., J. Am. Chem . Soc . 120:7916, 1998 참조)을 수행하거나 분자 역학(MD) 시뮬레이션을 시행함으로써 수득할 수 있다. 단백질의 MD 시뮬레이션은 컴퓨터에서 수행되고, 원자의 서로 간의 물리적 상호작용을 계산함으로써 임의의 기간 동안 모든 단백질 원자들의 운동을 결정하도록 한다. MD 시뮬레이션의 결과는 연구된 단백질의 시뮬레이션 기간 동안의 궤적이다. 궤적(또한 스냅샷으로도 불림)은 단백질 구조들의 총체이며, 시뮬레이션 기간 동안, 예컨대, 1 피코세컨드(ps)마다 주기적으로 수집된다. 스냅샷의 총체를 분석함으로써, 단백질 아미노산 잔기의 유연성을 정량할 수 있다는 것이다. 따라서, 유연한 잔기는 그 잔기가 존재하는 폴리펩티드의 맥락 내의 상이한 구조의 총체를 채택하는 잔기이다. MD 방법은 당해 분야에 공지되어 있으며, 예컨대, 문헌(Brooks et al. "Proteins: A Theoretical Perspective of Dynamics, Structure and Thermodynamics" (Wiley, New York, 1988)을 참조한다. Amber(Case et al. J. Comp. Chem. 26:1668, 2005; Brooks et al. J. Comp. Chem . 4:187, 1983; 및 MacKerell et al. (1998) "The Encyclopedia of Computational Chemistry" vol. 1:271-177, Schleyer et al., eds. Chichester: John Wiley & Sons 참조) 또는 Impact(Rizzo et al. J. Am. Chem . Soc . 122:12898, 2000 참조)와 같은 몇몇 소프트웨어들이 MD 시뮬레이션을 가능하게 한다.
대부분의 단백질 복합체는 상대적으로 크고 평면인 매복 표면을 공유하고, 결합 파트너의 유연성이 그들을 서로 구조적으로 맞출 수 있게 하는 그들의 가소성의 기원을 제공한다(Sundberg and Mariuzza, Structure 8, R137-R142, 2000). 이처럼, "유도 맞춤”의 예가 단백질 계면에서 지배적인 역할을 수행하는 것으로 나타난다. 또한, 단백질이 실제로 다양한 형태 크기 및 조성의 리간드에 결합하고(Protein Science 11:184-187, 2002) 구조적 다양성이 상이한 파트너를 인식하는 능력의 필수적인 구성요소인 것으로 보인다(James et al., Science 299:1362, 2003)는 것을 나타내는 데이터가 꾸준히 증가하고 있다. 유연한 잔기는 단백질-단백질 파트너의 결합에 관련된다(Grunberg et al., Structure 14, 683, 2006).
유연성 잔기들은 상호작용 영역들의 총체를 제공하는 다양한 구조를 채택할 수 있고, 다양한 구조는 기억 B 세포에 의해 인식되고 면역 반응을 유발할 수 있다. 따라서, 항체는 구조형성부로부터의 다수의 잔기들을 변형함으로써 인간화될 수 있어서, 변형된 항체의 의해 제시되는 총체적인 구조 및 인식 영역이 인간 항체에 의해 채택된 총체적인 구조 및 인식 영역과 가능한 한 닮게 된다. 이는 제한된 수의 잔기들을 다음에 의해 변형함으로써 달성할 수 있다: (1) 모 mAb의 상동성 모델 구축 및 MD 시뮬레이션 수행; (2) 유연성 잔기의 분석 및 비인간 항체 분자의 가장 유연한 잔기의 식별 및 이종성 또는 분해 반응의 원천이 될 수 있는 잔기 또는 모티프의 식별; (3) 모항체와 가장 유사한 총체적인 인식 면적을 제시하는 인간 항체의 식별; (4) 돌연변이될 유연성 잔기의 결정, 이종성 및 분해의 원천이 될 수 있는 잔기 또는 모티프도 또한 돌연변이화된다; 및 (5) 공지된 T 세포 또는 B 세포 에피토프의 존재에 대한 검증. 유연성 잔기는, 본원에 교시된 바와 같이, 시뮬레이션 기간 동안 단백질 원자들과 물 용매의 상호작용을 고려한 내재 용매 모델을 사용한 MD 계산을 통해 밝혀질 수 있다.
일단 가변 경쇄 및 중쇄 내 유연성 잔기들의 세트가 식별되면, 관심있는 항체의 중쇄 및 경쇄 가변 영역 구조형성부와 밀접하게 닮은 인간 중쇄 및 경쇄 가변 영역 구조형성부의 세트가 식별된다. 이는, 예를 들어, 항체 인간 생식계열 서열의 데이터베이스에 대한 유연성 잔기들의 세트의 BLAST 검색을 사용하여 수행될 수 있다. 또한, 이는 모 mAb의 역학을 생식계열 표준구조의 라이브러리의 역학과 비교함으로써 수행될 수 있다. 항원에 대한 높은 친화도를 보존하기 위해 CDR 잔기 및 이웃하는 잔기는 검색으로부터 제외된다. 유연성 잔기들은 그런 다음 교체된다.
몇몇 인간 잔기들이 유사한 상동성을 나타낼 때, 선택은 또한 인간화 항체의 용액 거동에 영향을 미칠 수 있는 잔기들의 성질에 의해 이루어진다. 예를 들어, 소수성 잔기에 걸쳐 노출된 유연성 루프 내에는 극성 잔기들이 종종 나타날 것이다. 불안정성 및 이종성의 잠재적인 원천인 잔기들은 심지어 잔기들이 CDR에서 발견되더라도 또한 돌연변이화된다. 돌연변이는, 산소 라디칼, Asp-Pro 디펩티드의 산 불안정성 결합과 같은 산 불안정성 결합(Drug Dev . Res. 61:137, 2004)의 가수분해, 작은 아미노산(예를 들면, Gly, Ser, Ala, His, Asn 또는 Cys(J. Chromatog . 837:35, 2006))이 뒤따르는 노출된 아스파라긴 잔기와 함께 발견되는 탈아미드화 자리, 및 N-당화 자리(예를 들면, Asn-X-Ser/Thr 자리)로부터 기인할 수 있는 술폭사이드 형성에 따른 메티오닌의 노출을 포함할 것이다. 통상적으로, 노출된 메티오닌은 Leu에 의해 치환될 것이거나, 노출된 아스파라긴은 글루타민 또는 아스파르산염에 의해 교체될 것이거나, 후속 잔기가 변할 것이다. 당화 자리(Asn-X-Ser/Thr)에 대해서는, Asn 또는 Ser/Thr 잔기 중 하나가 변할 것이다.
얻어진 복합 항체 서열은 공지된 B 세포 또는 선형 T 세포 에피토프의 존재에 대하여 점검된다. 검색은, 예를 들어, 열람가능한 면역 에피토프 데이터베이스(Immune Epitope Data Base, IEDB)(PLos Biol. (2005) 3(3)e91)를 사용하여 수행할 수 있다. 만약 공지된 에피토프가 복합 서열 내에서 발견된다면, 다른 세트의 인간 서열을 검색하고 치환할 수 있다. 따라서, 미국 특허 제5,639,641호의 표면치환법과는 다르게, B 세포 매개 및 T 세포 매개 면역원성 반응이 모두 본 방법에 의해 다루어질 수 있다. 본 방법은, 또한, CDR 이식에서 때때로 관찰되는 활성의 소실 문제를 회피한다(미국 특허 제5,530,101호). 또한, 안정성 및 용해성 문제들도 조작 및 선택 과정에서 고려되어, 낮은 면역원성, 높은 항원 친화도, 및 개선된 생물물리학적 성질을 위해 최적화된 항체가 얻어진다.
일부 구현예에서, 탈면역화가 항체 또는 이의 항원 결합 단편의 면역원성을 낮추기 위해 사용될 수 있다. 본원에서 사용된, "탈면역화”라는 용어는 T 세포 에피토프를 변형하기 위한 항체 또는 이의 항원 결합 단편의 변경을 포함한다(예컨대, 국제 공개 번호 WO9852976A1, WO0034317A2를 참조). 예를 들어, 출발 항체로부터의 VH 및 VL 서열이 분석될 수 있고, 상보성 결정 영역(CDR) 및 서열 내 다른 주요 잔기들과 비교하여 에피토프의 위치를 보여주는 인간 T 세포 에피토프 "지도”가 각각의 V 영역으로부터 생성될 수 있다. T 세포 에피토프 지도로부터의 개별적인 T 세포 에피토프들이 최종 항체의 활성을 변경시킬 위험이 낮은 대안적인 아미노산 치환을 식별하기 위해 분석된다. 아미노산 치환의 조합을 포함하는 다양한 대안적인 VH 및 VL 서열들이 설계되고, 이러한 서열들은, 이어서, 본원에 개시된 진단 및 치료 방법에 사용하기 위해 다양한 PAI-1 특이적 항체 또는 이의 단편에 삽입된 다음, 기능에 대해 시험된다. 통상적으로, 12 및 24개 사이의 변이 항체들이 생성되고 시험된다. 다음으로, 변형된 V 및 인간 C 영역을 포함하는 완전한 중쇄 및 경쇄 유전자가 발현 벡터 내로 클로닝되고, 이어서 플라스미드가 전항체의 생성을 위해 세포주 내로 도입된다. 그런 다음, 항체들은 적절한 생화학 및 생물학적 검정으로 비교되고, 최적의 변이체가 식별된다.
ii) 효과기 기능 및
Fc
변형
본원에 개시된 항-PAI-1 항체는 하나 이상의 효과기 기능을 매개하는 항체 불변 영역(예컨대, IgG 불변 영역, 예컨대, 인간 IgG 불변 영역, 예컨대, 인간 IgG1 또는 IgG4 불변 영역)을 포함할 수 있다. 예를 들어, 항체 불변 영역과 보체의 C1 구성요소의 결합은 보체 시스템을 활성화시킬 수 있다. 보체의 활성화는 세포 병원체의 옵소닌화 및 용해에 중요하다. 보체의 활성화는 또한 염증성 반응을 자극하고 또한 자가면역 과민증과 관련될 수 있다. 또한, 항체는 세포 상의 Fc 수용체(FcR)에 결합하는 항체 Fc 영역 상의 Fc 수용체 결합 자리를 가지는 Fc 영역을 통하여 다양한 세포 상의 수용체에 결합한다. IgG(감마 수용체), IgE(엡실론 수용체), IgA(알파 수용체) 및 IgM(뮤 수용체)을 포함하는 상이한 클래스의 항체에 특이적인 다수의 Fc 수용체가 존재한다. 세포 표면 상의 항체 Fc 수용체에 대한 항체의 결합은 항체로 코팅된 입자의 포획 및 파괴, 면역 복합체들의 제거, 살상 세포들에 의한 항체로 코팅된 표적 세포들의 용해(항체 의존성 세포 매개 세포독성, 또는 ADCC로 불림), 염증성 매개체들의 방출, 태반 이동 및 면역글로불린 생성의 제어를 포함하는 다수의 중요하고 다양한 생물학적 반응들을 유발한다. 일정한 구현예에서, 본원에 개시된 항체 또는 이의 단편은 Fc 감마 수용체에 결합한다. 대안적인 구현예에서, 본원에 개시된 항-PAI-1 항체는 하나 이상의 효과기 기능(예컨대, ADCC 활성)이 없는 불변 영역을 포함할 수 있거나 Fc 수용체에 결합할 수 없다.
본원에 개시된 일정한 구현예는 하나 이상의 불변 영역 도메인의 적어도 하나의 아미노산이 결실되거나 그렇지 않으면 변경되어 원하는 생화학적 특성(예를 들면, 대략 동일한 면역원성의 미변경 전항체와 비교할 때, 감소되거나 향상된 효과기 기능, 비공유적으로 이량체화되는 능력, 신체 내 특정 자리(예를 들어, 종양의 자리, 또는 특정 기관)로 국소화되는 능력의 증가, 감소된 혈청 반감기, 또는 증가된 혈청 반감기)을 제공하는 항-PAI-1항체를 포함한다. 예를 들어, 본원에 개시된 진단 및 치료 방법에 사용하기 위한 일정 항체 또는 이의 단편은 도메인이 결실된 항체로, 면역글로불린 중쇄와 유사한 폴리펩티드 사슬을 포함하지만, 하나 이상의 중쇄 도메인의 적어도 하나의 부위가 결여되어 있다. 예를 들어, 일정 항체에서, 변형된 항체의 불변 영역의 하나의 전체 도메인이 결실될 것이고, 예를 들어, CH2 도메인의 전부 또는 일부가 결실될 것이다.
다른 일정 구현예에서, 항-PAI-1 항체는 상이한 항체 동형으로부터 유래한 불변 영역(예컨대, 인간 IgG1, IgG2, IgG3, 또는 IgG4 중 2종 이상으로부터 유래한 불변 영역)을 포함한다. 다른 구현예에서, 항-PAI-1 항체는 키메라 힌지(즉, 상이한 항체 동형의 힌지 도메인으로부터 유래한 힌지 부위, 예컨대 IgG4 분자로부터의 상부 힌지 도메인 및 IgG1 중간 힌지 도메인을 포함하는 힌지)를 포함한다. 일 구현예에서, 항-PAI-1 항체는 인간 IgG4 분자로부터의 Fc 영역 또는 이의 부위 및 분자의 코어 힌지 영역 내 Ser228Pro 돌연변이(카바트 번호부여)를 포함한다.
일정한 항-PAI-1 항체에서, Fc 부위는 효과기 기능을 높이거나 낮추기 위해 당해 분야에 공지된 기술을 사용하여 돌연변이화될 수 있다. 예를 들어, 불변 영역 도메인의 (점돌연변이 또는 다른 수단을 통한) 결실 및 불활성화는 변형된 혈중 항체의 Fc 수용체 결합을 감소시킴으로써 종양 국소화를 증가시킨다. 다른 경우에서, 본 발명과 일치하는 불변 영역 변형은 보체 결합을 약화시켜서, 접합된 세포독소의 혈청 반감기 및 비특이적 결합을 감소시키는 것일 수 있다. 이황화 결합 또는 올리고당 모이어티를 변형시키기 위해 불변 영역의 여전히 다른 변형이 사용될 수 있고, 증가된 항원 특이성 또는 유연성으로 인해 국소화를 향상시킬 수 있다. 변형의 결과적인 생리학적 프로파일, 생물학적 이용가능성 및 다른 생화학적 효과, 예를 들면, 종양 국소화, 생물학적 분배, 및 혈청 반감기는 불필요한 실험 없이 잘 알려진 면역학적 기술들을 사용하여 쉽게 측정되고 정량될 수 있다.
일정 구현예에서, 본원에 개시된 항체에 채용된 Fc 도메인은 Fc 변이체이다. 본원에서 사용된 "Fc 변이체”라는 용어는 상기 Fc 도메인이 유래된 야생형 Fc 도메인과 비교하여 적어도 하나의 아미노산 치환을 가지는 Fc 도메인을 지칭한다. 예를 들어, Fc 도메인이 인간 IgG1 항체로부터 유래한 경우, 상기 인간 IgG1 Fc 도메인의 Fc 변이체는 상기 Fc 도메인과 비교하여 적어도 하나의 아미노산 치환을 포함한다.
Fc 변이체의 아미노산 치환(들)은 Fc 도메인 내 임의의 위치(즉, 임의의 EU 관례 아미노산 위치)에 위치할 수 있다. 일 구현예에서, Fc 변이체는 힌지 도메인 또는 이의 부위에 위치한 아미노산 위치에 치환을 포함한다. 다른 구현예에서, Fc 변이체는 CH2 도메인 또는 이의 부위에 위치한 아미노산 위치에 치환을 포함한다. 다른 구현예에서, Fc 변이체는 CH3 도메인 또는 이의 부분에 위치한 아미노산 위치에 치환을 포함한다. 다른 구현예에서, Fc 변이체는 CH4 도메인 또는 이의 부분에 위치한 아미노산 위치에 치환을 포함한다.
본원에 개시된 항체는 효과기 기능 또는 FcR 결합을 개선(예컨대, 감소 또는 향상)하는 것으로 알려진 임의의 당해 분야에 알려진 Fc 변이체를 채용할 수 있다. 상기 Fc 변이체는, 예를 들어, 각각 참조로서 본원에 포함된 국제 PCT 공개 WO88/07089A1, WO96/14339A1, WO98/05787A1, WO98/23289A1, WO99/51642A1, WO99/58572A1, WO00/09560A2, WO00/32767A1, WO00/42072A2, WO02/44215A2, WO02/060919A2, WO03/074569A2, WO04/016750A2, WO04/029207A2, WO04/035752A2, WO04/063351A2, WO04/074455A2, WO04/099249A2, WO05/040217A2, WO05/070963A1, WO05/077981A2, WO05/092925A2, WO05/123780A2, WO06/019447A1, WO06/047350A2, 및 WO06/085967A2 또는 미국 특허 제5,648,260호; 제5,739,277호; 제5,834,250호; 제5,869,046호; 제6,096,871호; 제6,121,022호; 제6,194,551호; 제6,242,195호; 제6,277,375호; 제6,528,624호; 제6,538,124호; 제6,737,056호; 제6,821,505호; 제6,998,253호; 및 제7,083,784호에 개시된 아미노산 치환 중 임의의 하나를 포함할 수 있다. 예시적인 일 구현예에서, 본원에 개시된 항체는 EU 위치 268에 아미노산 치환을 포함하는 Fc 변이체(예컨대, H268D 또는 H268E)를 포함할 수 있다. 다른 예시적인 구현예에서, 본원에 개시된 항체는 EU 위치 239번(예컨대, S239D 또는 S239E) 또는 EU 위치 332번(예컨대, I332D 또는 I332Q)에 아미노산 치환을 포함할 수 있다.
일정 구현예에서, 본원에 개시된 항체는 항체의 항원 비의존성 효과기 기능, 특히 항체의 순환 반감기를 변경하는 아미노산 치환을 포함하는 Fc 변이체를 포함할 수 있다. 이러한 항체는 이러한 치환이 결여된 항체에 비교하여 증가하거나 감소된 FcRn에 대한 결합을 나타내며, 따라서, 혈청 내에서 각각 증가하거나 감소된 반감기를 가진다. FcRn에 대한 개선된 친화도를 가지는 Fc 변이체는 더 긴 혈청 반감기를 가지는 것으로 예상되며, 이러한 분자들은 투여되는 항체의 반감기가 긴 것이 바람직한 포유동물의 치료, 예컨대, 만성 질환 또는 장애를 치료하기 위한 방법에 유용하게 적용된다. 반대로, 감소된 FcRn 결합 친화도를 가지는 Fc 변이체는 더 짧은 반감기를 가지는 것으로 예상되며, 이러한 분자들은 또한, 예를 들어, 단축된 순환 시간이 유리할 수 있는 포유동물에의 투여, 예컨대 생체 내 진단 영상화 또는 출발 항체가 연장된 기간 동안 순환계에 존재하는 경우 독성 부작용을 가지는 상황에 유용하다. 감소된 FcRn 결합 친화도를 가지는 Fc 변이체는 또한 태반을 가로지르는 경향이 낮기 때문에 임신한 여성의 질환 또는 장애의 치료에 또한 유용하다. 추가로, 감소된 FcRn 결합 친화도가 바람직할 수 있는 다른 적용예는 뇌, 신장 또는 간으로의 국소화가 바람직한 적용예를 포함한다. 예시적인 일 구현예에서, 본원에 개시된 변경된 항체는 혈관으로부터 신장 사구체의 표피를 가로지르는 이동의 감소를 나타낸다. 다른 구현예에서, 본원에 개시된 변경된 항체는 뇌로부터 혈관 공간으로 뇌혈관 장벽(BBB)을 가로지르는 이동의 감소를 나타낸다. 일 구현예에서, 변경된 FcRn 결합을 가지는 항체는 Fc 도메인의 "FcRn 결합 루프” 내에 하나 이상의 아미노산 치환을 가지는 Fc 도메인을 포함한다. FcRn 결합 루프는 아미노산 잔기 280 내지 299(카바트 번호부여에 따름)로 구성된다. FcRn 결합 활성을 바꾸는 예시적인 아미노산 치환이 본원에 참조로서 포함된 국제 PCT 공개 WO05/047327에 개시되어 있다. 예시적인 일정 구현예에서, 본원에 개시된 항체 또는 이의 단편은 하기 치환 중 하나 이상을 포함하는 Fc 도메인을 포함한다: V284E, H285E, N286D, K290E 및 S304D(카바트 번호부여).
다른 구현예에서, 본원에 기술된 진단 및 치료 방법에 사용하기 위한 항체는 당화를 감소시키거나 제거하도록 변경된 불변 영역, 예컨대 IgG1 또는 IgG4 중쇄 불변 영역을 가진다. 예를 들어, 본원에 개시된 항체는 또한 항체의 당화를 변경하는 아미노산 치환을 포함하는 Fc 변이체를 포함할 수 있다. 예를 들어, 상기 Fc 변이체는 감소된 당화(예컨대, N- 또는 O- 결합 당화)를 가질 수 있다. 예시적인 구현예에서, Fc 변이체는 보통 아미노산 위치 297번(EU 번호부여)에서 발견되는 N-결합 글리칸의 당화의 감소를 포함한다. 다른 구현예에서, 항체는 당화 모티브(예를 들어, 아미노산 서열 NXT 또는 NXS를 포함하는 N-결합 당화 모티프)에 가깝게 또는 당화 모티프 내에 아미노산 치환을 가진다. 특정 구현예에서, 항체는 아미노산 위치 228 또는 299번(EU 번호부여)에 아미노산 치환을 가지는 Fc 변이체를 포함한다. 더욱 특정한 구현예에서, 항체는 S228P 및 T299A 돌연변이(EU 번호부여)를 포함하는 IgG1 또는 IgG4 불변 영역을 포함한다.
감소되거나 변경된 당화를 부여하는 예시적인 아미노산 치환은 본원에 참조로서 포함된 국제 PCT 공개 WO05/018572에 개시되어 있다. 일정한 구현예에서, 본원에 개시된 항체 또는 이의 단편은 당화를 제거하기 위해 변형된다. 이러한 항체 또는 이의 단편은 "어글리(agly)" 항체 또는 이(예컨대, "어글리 항체”)의 단편으로 지칭될 수 있다. 이론에 얽매이지 않으면서, "어글리” 항체, 또는 이의 단편은 개선된 생체 내 안전성 및 안정성 프로파일을 가질 수 있는 것으로 여겨진다. 예시적인 어글리 항체 또는 이의 단편은 Fc 효과기 기능이 결여된 IgG4 항체의 무당화 Fc 영역을 포함함으로써 PAI-1을 발현하는 보통의 중요 기관에 대한 Fc 매개 독성의 가능성을 제거한다. 여전히 다른 구현예에서, 본원에 개시된 항체 또는 이의 단편은 변경된 글리칸을 포함한다. 예를 들어, 항체는 Fc 영역의 Asn297의 N-글리칸 상에 감소된 수의 푸코스 잔기를 가질 수 있다(즉, 무푸코실화된다). 다른 구현예에서, 항체는 Fc 영역의 Asn297의 N-글리칸 상에 변경된 수의 시알산 잔기를 가질 수 있다.
iii) 공유 부착
본원에 개시된 항-PAI-1 항체는, 예컨대 공유 부착이 항체가 그의 동족 에피토프에 특이적으로 결합하는 것을 방지하지 않도록 하는 항체에 대한 분자의 공유 부착에 의해 변형될 수 있다. 제한의 방식이 아니라, 예를 들어, 본원에 개시된 항체 또는 이의 단편은 당화, 아세틸화, 페길화, 인산화, 아미드화, 공지된 보호기/차단기에 의한 유도체화, 단백질 가수분해, 세포 리간드 또는 다른 단백질 등에 의한 결합에 의해 변형될 수 있다. 특이적 화학적 분해, 아세틸화, 포밀화 등을 포함하나 이에 제한되지는 않는 공지된 기술에 의해 다수의 화학적 변형 중 임의의 것이 수행될 수 있다. 추가적으로, 유도체는 하나 이상의 비 고전적 아미노산 치환을 포함할 수 있다.
본원에 개시된 항체 또는 이의 단편은 또한 N- 또는 C- 말단 이종 폴리펩티드에 재조합적으로 융합되거나 폴리펩티드 또는 다른 조성물에 화학적으로 접합(공유 및 비공유 접합을 포함함)될 수 있다. 예를 들어, 항-PAI-1 항체가 검출 검정에서 표지로서 유용한 분자 및 이종 폴리펩티드, 약물, 방사성핵종 또는 독소와 같은 효과기 분자에 재조합적으로 융합되거나 접합될 수 있다. 예컨대, 국제 PCT 공개 번호 WO 92/08495; WO 91/14438; WO 89/12624; 미국 특허 제5,314,995호; 및 EP 396,387을 참조한다.
항-PAI-1 항체가 생체 내 반감기를 증가시키거나 당해 분야에 공지된 방법을 사용하는 면역검정법에 사용하기 위해 이종 폴리펩티드에 융합될 수 있다. 예를 들어, 일 구현예에서, 생체 내 반감기를 증가시키기 위해 PEG가 본원에 개시된 항-PAI-1 항체에 접합될 수 있다(Leong, S. R., et al., Cytokine 16:106, 2001; Adv. in Drug Deliv. Rev. 54:531, 2002; 또는 Weir et al., Biochem. Soc. Transactions 30:512, 2002).
더욱이, 본원에 개시된 항-PAI-1 항체는 그들의 정제 또는 검출을 촉진하기 위한 펩티드와 같은 마커 서열에 융합될 수 있다. 일정한 구현예에서, 마커 아미노산 서열은 상업적으로 입수가능한 많은 다른 것들 중에서 pQE 벡터(QIAGEN, Inc., 9259 Eton Avenue, Chatsworth, Calif., 91311)에 제공되는 태그와 같은 헥사 히스티딘 펩티드이다. 문헌(Gentz et al., Proc. Natl. Acad. Sci. USA 86:821-824, 1989)에 기술된 바와 같이, 예를 들어, 헥사 히스티딘은 융합 단백질의 편리한 정제를 제공한다. 정제에 유용한 다른 펩티드 태그는, 이에 제한되지는 않지만, 인플루엔자 헤마글루티닌 단백질로부터 유래된 에피토프에 상응하는 "HA" 태그(Wilson et al., Cell 37:767, 1984) 및 "플래그” 태그를 포함한다.
본원에 개시된 항-PAI-1 항체는 비접합 형태로 사용될 수 있거나, 예컨대, 분자의 치료적 성질을 개선하거나, 표적 검출을 용이하게 하거나, 환자의 영상화 또는 치료를 위해 다양한 분자들 중 적어도 하나에 접합될 수 있다. 본원에 개시된 항-PAI-1 항체는, 정제가 수행될 때, 정제의 이전 또는 이후에 표지되거나 접합될 수 있다. 특히, 본원에 개시된 항-PAI-1 항체는 치료제, 전구약물, 펩티드, 단백질, 효소, 바이러스, 지질, 생물학적 반응 조절제, 약학적 제제 또는 PEG에 접합될 수 있다.
본 발명은 진단제 또는 치료제에 접합된 항-PAI-1 항체를 더 포함한다. 항-PAI-1 항체는 진단용으로 사용될 수 있는데, 예컨대, 주어진 치료 또는 예방 계획의 효능을 결정하기 위한 임상 시험 절차의 일부로서, 예를 들어, 면역 세포 장애(예컨대, CLL)의 진행 또는 발달을 감시하기 위해 사용될 수 있다. 검출은 항-PAI-1 항체를 검출가능한 물질에 연결시킴으로써 촉진될 수 있다. 검출가능한 물질의 예는 다양한 효소, 보결 원자단, 형광 물질, 발광 물질, 생물발광 물질, 방사성 물질, 다양한 양전자 방출 단층 촬영을 사용한 양전자 방출 금속, 및 비방사성 상자성 금속 이온들을 포함한다. 예를 들어, 본 발명에 따른 진단제로서 사용하기 위한 항체에 접합될 수 있는 금속 이온에 대한 미국 특허 제4,741,900호를 참조한다. 적합한 효소의 비제한적인 예는 홀스래디쉬 과산화효소, 알칼라인 포스파타제, 베타-갈락토시다제 또는 아세틸콜린에스테라제를 포함하고; 적합한 보결 원자단 복합체 비제한적인 예는 스트렙트아비딘/비오틴 및 아비딘/비오틴을 포함하고; 적합한 형광 물질의 비제한적인 예는 움벨리페논, 플루오레세인, 플루오레세인 이소티오시아네이트, 로다민, 디클로로트리아지닐라민 플루오레세인, 단실 클로라이드 또는 피코에리트린을 포함하고; 발광 물질의 비제한적인 예는 루미놀을 포함하고; 생물발광 물질의 비제한적인 예는 루시페라제, 루시페린, 및 에쿼린을 포함하고; 적합한 방사성 물질의 비제한적인 예는 125I, 131I, 111In 또는 99Tc을 포함한다.
본원에 개시된 진단 및 치료방법에 사용하기 위한 항-PAI-1 항체는 (방사성동위원소, 세포독성 약물 또는 독소와 같은) 세포독소 치료제, 세포정지제, 생물학적 독소, 전구약물, 펩티드, 단백질, 효소, 바이러스, 지질, 생물학적 반응 조절제, 약학적 제제, 면역학적으로 활성인 리간드(예컨대, 림포카인 또는 얻어진 분자가 신생 세포 및 T 세포와 같은 효과기 세포 둘 다에 결합하는 다른 항체), 또는 PEG에 접합될 수 있다.
다른 구현예에서, 본원에 개시된 진단 및 치료 방법에 사용하기 위한 항-PAI-1 항체는 종양 세포 생장을 낮추는 분자에 접합될 수 있다. 다른 구현예에서, 개시된 조성물은 약물 또는 전구약물에 연결된 항체 또는 이의 단편을 포함할 수 있다. 본원에 개시된 여전히 다른 구현예는 리신(ricin), 겔로닌(gelonin), 슈도모나스 외독소 또는 디프테리아 독소와 같은 특이적인 생물 독소 또는 그들의 세포독성 단편에 접합된 항체 또는 이의 단편의 용도를 포함한다. 접합 또는 미접합 항체 사용의 선택은 암의 유형 및 단계, 부가 치료(예컨대, 화학요법 또는 외부방사선)의 사용 및 환자 병태에 의존할 것이다. 본원의 교시를 고려하여 쉽게 이러한 선택을 내릴 수 있다는 것이 이해될 것이다.
이전의 연구에서, 동위원소로 표지된 항 종양 항체가 동일 모델, 및 일부 경우 인간에서 종양 세포를 파괴하기 위해 성공적으로 사용되어 왔다는 것이 이해될 것이다. 예시적인 방사성동위원소는 90Y, 125I, 131I, 123I, 111In, 105Rh, 153Sm, 67Cu, 67Ga, 166Ho, 177Lu, 186Re 및 188Re를 포함한다. 방사성 핵종은 핵 DNA에서 세포 사멸을 초래하는 다수의 가닥 끊김을 유발하는 전리 방사선을 생성함으로써 작용한다. 치료용 접합체를 생성하기 위해 사용된 동위원소는 경로 길이가 짧은 통상적으로 고에너지 알파 또는 베타 입자를 생성한다. 이러한 방사성 핵종은 그들과 밀접하게 인접한 세포, 예를 들어 접합체가 부착되거나 도입된 신생 세포를 살상한다. 그들은 비 국소화 세포에 거의 영향을 미치지 않거나 전혀 영향을 미치지 않는다. 방사성핵종은 필수적으로 비면역원성이다.
IV. 항-
PAI
-1 항체 또는 이의 항원 결합 단편의 발현
위에 기재된 바와 같은 본원에 개시된 항-PAI-1 항체를 제공하기 위해 단리된 유전 물질의 조작 후, 통상적으로, 원하는 양의 청구되는 항체 또는 이의 단편을 생성하기 위해 사용될 수 있는 숙주 세포에 도입하기 위한 발현 벡터 내에 유전자를 삽입한다.
다른 구현예에서, 본원에 개시된 항-PAI-1 항체 또는 이의 단편은 폴리시스트론 구조체를 통해 발현될 수 있다. 이러한 발현 시스템에서, 항체의 중쇄 및 경쇄와 같은 관심있는 다수의 유전자 산물은 단일 폴리시스트론 컨스트럭트로부터 생성될 수 있다. 이러한 시스템은 유리하게는 진핵 숙주 세포 내에서 상대적으로 높은 수준의 본원에 개시된 폴리펩티드를 제공하기 위해 내부 리보솜 유입 자리(internal ribosome entry site, IRES)를 사용한다. 친화성의 IRES 서열이 본원에 참조로서 포함된 미국 특허 제6,193,980호에 개시되어 있다. 당업자는 이러한 발현 시스템이 본 출원에 개시된 폴리펩티드의 전체 범위를 효과적으로 생성하기 위해 사용될 수 있다는 것을 이해할 것이다.
일 구현예에서, 항체 발현을 위해 사용되는 숙주 세포주는 포유동물 기원이고; 당업자는 그 안에서 발현될 원하는 유전자 생성물에 가장 적합한 특정 숙주 세포주를 결정할 수 있다. 예시적인 숙주 세포는, 이에 제한되지는 않으나, DG44 및 DUXB11(차이니즈 햄스터 난소주, DHFR 음성), HELA(인간 경부암종), CVI(원숭이 신장주), COS(SV40 T 항원을 가지는 CVI 의 유도체), R1610(차이니즈 햄스터 섬유아세포) BALBC/3T3(마우스 섬유아세포), HAK(햄스터 신장주), SP2/O(마우스 골수종), BFA-1c1BPT(소 내피 세포), RAJI(인간 림프구), 293(인간 신장)을 포함한다. 일 구현예에서, 세포주는 항체가 발현된 세포주(예컨대, PER.C6.RTM.(Crucell) 또는 FUT8-녹아웃 CHO 세포주(Potelligent.RTM. 세포)(Biowa, Princeton, N.J.)로부터 변경된 당화, 예컨대, 무푸코실화를 제공한다. 일 구현예에서, NS0 세포가 사용될 수 있다. CHO 세포가 일정한 특정 구현예에서 사용될 수 있다. 숙주 세포주는 통상적으로 상업적인 서비스인 미국 세포주 은행(American Tissue Culture Collection) 또는 공개된 문헌으로부터 입수가능하다.
시험관 내 생성은 대량의 원하는 폴리펩티드를 제공하는 스케일 업을 가능하게 한다. 조직 배양 조건 하에 포유동물 세포 배양을 위한 기술들이 당해 분야에 공지되어 있고, 예컨대, 공기양수 반응기 또는 연속 교반 반응기 내에서의 균일 현탁 배양, 또는, 예컨대, 중공 섬유, 미세 캡슐 내, 아가로스 마이크로비드 또는 세라믹 카트리지 상에서의 고정되거나 포획된 세포 배양을 포함한다. 필요하거나 원한다면, 폴리펩티드의 용액은 통상적인 크로마토그래피 방법, 예를 들어, 겔 여과, 이온 교환 크로마토그래피, DEAE-셀룰로오스를 사용한 크로마토그래피 또는 (면역) 친화도 크로마토그래피에 의해 정제될 수 있다.
본원에 개시된 항-PAI-1 항체, 또는 이의 단편을 암호화하는 유전자는 또한 박테리아 또는 효모 또는 식물 세포와 같은 비 포유동물 세포에서 발현될 수 있다. 이러한 관점에서, 다양한 단세포 비포유동물 미생물(예를 들면, 박테리아), 즉, 배양물에서 생장할 수 있거나 발효될 수 있는 미생물이 또한 형질전환될 수 있다는 것이 이해될 것이다. 형질전환이 잘 되는 박테리아는 장내세균과(예를 들면, 에스체리키아 콜라이 또는 살모넬라 균주); 간균과(예를 들면, 바실러스 서브틸리스); 뉴모코커스; 스트렙토코커스, 및 헤모필러스 인플루엔자의 구성원을 포함한다. 또한, 박테리아에서 발현될 때, 폴리펩티드는 봉입소체의 일부가 될 수 있는 것으로 이해될 것이다. 폴리펩티드는 단리되고, 정제된 다음 기능적 분자 내로 조립되어야 한다.
원핵생물에 더하여, 진핵 미생물도 또한 사용될 수 있다. 다수의 다른 균주들도 흔히 사용가능하지만, 사카로마이세스 세레비지에 또는 통상적인 빵 효모가 진핵 미생물 중에서 가장 흔하게 사용된다. 사카로마이세스 내에서의 발현을 위해, 예를 들어, 플라스미드 YRp7(Stinchcomb et al., Nature, 282:39 (1979); Kingsman et al., Gene, 7:141 (1979); Tschemper et al., Gene, 10:157 (1980))이 흔히 사용된다. 이러한 플라스미드는 이미 트립토판에서 생장하는 능력이 결여된 효모 돌연변이 균주(예를 들어, ATCC No. 44076 또는 PEP4-1(Jones, Genetics, 85:12 (1977))에 대한 선택 마커를 제공하는 TRP1 유전자를 함유하고 있다. 그런 다음, 효모 숙주 세포 게놈의 특성으로서 trpl 손상의 존재는 트립토판의 부재 하에서의 생장에 의해 형질전환을 검출하기 위한 효과적인 환경을 제공한다.
V. 항-PAI-1 항체의 투여 방법 및 약학적 제제.
다른 양태에서, 본 발명은 항-PAI-1 항체 또는 이의 단편을 포함하는 약학적 조성물을 제공한다.
본원에 개시된 항체 또는 이의 단편을 제조하고 대상체에 투여하는 방법은 당업자에게 잘 알려져 있거나 당업자에 의해 용이하게 결정된다. 본원에 개시된 항체 또는 이의 단편의 투여 경로는 경구, 흡입 또는 국소에 의한 비경구일 수 있다. 본원에서 사용된 비경구라는 용어는 정맥내, 동맥내, 복강내, 근육내, 피하, 직장 또는 질 투여를 포함한다. 비경구 투여의 정맥내, 동맥내, 피하 및 근육내 형태가 일정한 구현예에서 사용될 수 있다. 이러한 투여 형태들 모두가 명백하게 본원에 개시된 범주 내인 것으로 고려되지만, 투여를 위한 형태는 주사용 용액, 특히 정맥 내 또는 동맥 내 주사 또는 점적주입일 수 있다. 보통, 주사에 적합한 약학적 조성물은 완충액(예컨대, 아세테이트, 인산염, 또는 시트르산 완충액), 계면활성제(예컨대, 폴리소르베이트), 선택적으로 안정화제(예컨대, 인간 알부민)등을 포함할 수 있다. 그러나, 본원의 교시와 맞는 다른 방법들에서, 폴리펩티드는 유해한 세포 군집의 자리에 직접적으로 전달됨으로써 질환 조직을 치료제에 노출하는 것을 증가시킬 수 있다.
비경구 투여를 위한 제제는 멸균 수용액 또는 비수용액, 현탁액 및 에멀젼을 포함한다. 비수용성 용매의 예는 프로필렌 글리콜, 폴리에틸렌 글리콜, 올리브 오일과 같은 식물성 오일 및 에틸 올레이트와 같은 주사용 유기 에스테르이다. 수용성 담체는, 염분 및 완충 배지를 포함하는, 물, 알코올성/수성 용액, 살린 에멀젼 또는 현탁액을 포함한다. 대상 발명에서, 약학적으로 허용가능한 담체는, 이에 제한되지는 않지만, 0.01 내지 0.1 M, (예를 들어, 0.05 M) 인산 완충액 또는 0.8% 염수를 포함한다. 다른 흔한 비경구 비히클은 인산 나트륨 용액, 링거 덱스트로스(Ringer's dextrose), 덱스트로스 및 염화 나트륨, 젖산화 링거액(Ringer's), 또는 고정유를 포함한다. 정맥 내 비히클은 링거 덱스트로스에 기반한 것 등과 같은 전해질 보충물, 유체 및 영양 보충물 등을 포함한다. 예를 들어, 항미생물제, 항산화제, 킬레이팅제 및 불활성화 기체 등과 같은 보존제 및 기타 첨가제들이 또한 존재할 수 있다. 더욱 상세하게는, 주사용으로 적합한 약학적 조성물은 멸균 수용액(수용성) 또는 분산액 및 멸균 주사 용액 또는 분산액의 즉석 조제를 위한 멸균 분말을 포함한다. 이러한 경우에, 조성물은 반드시 멸균되어야 하고, 용이한 주사가능성이 존재하는 정도의 유체여야 한다. 조성물은 제조 및 저장의 조건 하에 안정해야 하고, 일정한 구현예에서는 박테리아 및 진균류와 같은 미생물의 오염 작용으로부터 보존될 것이다. 담체는, 예를 들어, 물, 에탄올, 폴리올(예컨대, 글리세롤, 프로필렌 글리콜, 및 액체 폴리에틸렌 글리콜 등) 및 이의 적합한 혼합물을 함유하는 용매 또는 분산 매질일 수 있다. 예를 들어, 레시틴과 같은 코팅의 사용, 분산의 경우에서 요구되는 입자 크기의 유지, 및 계면활성제의 사용에 의해 적절한 유동성이 유지될 수 있다. 미생물의 작용의 방지는, 예를 들어, 파라벤, 클로로부탄올, 페놀, 아스코르브산, 및 티메로살 등을 포함하는 다양한 항박테리아 및 항진균제에 의해 달성될 수 있다. 일정한 구현예에서, 등장제, 예를 들어, 자당, 폴리알코올(예를 들면, 만니톨, 소르비톨), 또는 염화 나트륨이 조성물 내에 포함된다. 주사용 조성물의 흡수 연장은 흡수를 지연시키는 제제, 예를 들어, 알루미늄 모노스테아레이트 및 젤라틴을 조성물 내에 포함시킴으로써 야기될 수 있다.
임의의 경우, 멸균 주사 용액은 활성 화합물(예컨대, 항체 그 자체 또는 다른 활성제와의 조합)을 요구되는 양으로 요구에 따라 본원에 나열된 성분 중 하나 또는 조합을 가지는 적절한 용매에 포함시키고 여과 멸균함으로써 제조될 수 있다. 일반적으로, 분산액은 활성 화합물을 염기 분산 매질 및 위에 나열된 성분으로부터 요구되는 기타 성분들을 함유하는 멸균 비히클에 포함시킴으로써 제조될 수 있다. 멸균 주사 용액의 제조를 위한 멸균 분말의 경우, 제조 방법은 사전 멸군 여과 용액으로부터의 임의의 추가적인 원하는 성분에 더해진 유효 성분의 분말을 산출하는 진공 건조 및 동결 건조일 수 있다. 당해 분야에 공지된 방법에 따라 주사를 위한 제제가 가공되고, 앰플, 백, 병, 주사기 또는 바이알과 같은 용기에 채워지고, 무균 상태 하에 밀봉된다. 또한, 제제는 각각이 참조로서 본원에 포함된 동시 계류중인 미국 일련번호 제09/259,337호 및 미국 일련번호 제09/259,338호에 기술된 키트와 같은 키트의 형태 내에 포장되고 판매될 수 있다. 이러한 제조 물품은, 일 구현예에서, 연관된 조성물이 자가면역 또는 종양 장애로 고통받는 대상체를 치료하거나 자가면역 또는 종양 장애의 예후의 표지로 유용하다는 것을 나타내는 표지 또는 포장 인서트를 가질 것이다.
전술된 병태를 치료하기 위한 본원에 개시된 안정화된 항체 또는 이의 단편의 유효 용량은 투여의 수단, 표적 자리, 환자의 생리학적 상태, 환자가 인간인지 또는 동물인지의 여부, 투여되는 다른 약제들, 및 치료가 예방적인지 또는 치료적인지의 여부를 포함하는 많은 다른 인자들에 따라 달라진다. 대개, 환자는 인간이지만, 유전자이식 포유동물을 포함하는 인간이 아닌 포유동물들도 또한 치료될 수 있다. 안전성 및 효능을 최적화하기 위해, 당업자에게 공지된 통상적인 방법을 사용하여 치료 투여량이 적정될 수 있다.
본원에 개시된 항체로의 수동 면역에서, 투여량은, 예컨대, 숙주 체중의 약 0.0001 내지 100 mg/kg, 더욱 흔하게는 0.01 내지 5 mg/kg(예컨대, 0.02 mg/kg, 0.25 mg/kg, 0.5 mg/kg, 0.75 mg/kg, 1 mg/kg, 2 mg/kg 등)의 범위일 수 있다. 예를 들어, 투여량은 체중 1 kg 당 1 mg 또는 체중 1 kg 당 10 mg 또는 1 내지 10 mg/kg의 범위, 또는 특정한 구현예에서, 적어도 1 mg/kg일 수 있다. 위의 범위의 중간의 용량들도 또한 본원에 개시된 범주 내에 포함되는 것으로 의도된다.
대상체는 이러한 용량을 매일, 격일, 매주 또는 실험적 분석에 의해 결정된 임의의 다른 일정에 따라 투여받는다. 하나의 예시적인 치료는 연장된 기간, 예를 들어, 적어도 6개월에 걸친 다수의 투여량으로의 투여를 수반한다. 추가적인 예시적인 치료 계획은 2주마다 1회 또는 매월 1회 또는 3 내지 6 개월마다 1회의 투여를 수반한다. 예시적인 투여 일정은 매일 1 내지 10 mg/kg 또는 15 mg/kg, 격일로 30 mg/kg 또는 매주 60 mg/kg를 포함한다. 일부 방법에서, 상이한 결합 특이성을 가지는 2종 이상의 단일클론 항체가 동시에 투여되고, 이 경우 투여되는 각 항체의 투여량은 지시된 범위 내이다.
본원에 개시된 항체 또는 이의 단편은 다수 회로 투여될 수 있다. 단일 투여량 사이의 간격은, 예컨대, 매일, 주간, 월간 또는 연간이 될 수 있다. 간격은 또한 환자에서 폴리펩티드 또는 표적 분자의 혈중 수준을 측정함으로써 지시되는 바와 같이 불규칙할 수 있다. 일부 방법에서, 투여량은 일정한 혈장 항체 또는 독소 농도, 예컨대, 1 내지 1000 ug/ml 또는 25 내지 300 ug/ml을 달성하기 위해 조정된다. 대안적으로, 항체 또는 이의 단편은 서방 제제로 투여될 수 있고, 이 경우 더 적은 빈도의 투여가 요구된다. 투여량 및 빈도는 환자 내 항체의 반감기에 따라 달라진다. 일반적으로, 인간화 항체는 더 긴 반감기를 나타내고, 다음으로 키메라 항체 및 비인간 항체가 뒤따른다. 일 구현예에서, 본원에 개시된 항체 또는 이의 단편은 미접합 형태로 투여될 수 있다. 다른 구현예에서, 본원에 개시된 항체는 접합 형태로 다수회 투여될 수 있다. 여전히 다른 구현예에서, 본원에 개시된 항체 또는 이의 단편은 미접합 형태로 투여된 다음, 접합된 형태로 투여되거나 이 반대일 수 있다.
투여의 빈도 및 투여량은 치료가 예방적인지 또는 치료적인지에 따라 달라질 수 있다. 예방적인 적용에서, 본 항체 또는 이의 칵테일을 함유하는 조성물은 환자의 저항성을 향상시키기 위해 아직 질병상태가 아닌 환자에게 투여된다. 이러한 양은 "예방적 유효량”으로 정의된다. 이러한 용도에서, 정확한 양은, 다시, 환자의 건강 상태 및 일반적인 면역에 의존하지만, 일반적으로 용량 당 0.1 내지 25 mg, 특히 용량 당 0.5 내지 2.5 mg의 범위이다. 긴 기간에 걸쳐서 상대적으로 낮은 투여량이 상대적으로 덜 잦은 간격으로 투여될 수 있다. 일부 환자는 그들의 남은 일생 동안 치료를 받는 것을 계속한다.
치료적 적용에서, 질환의 진행이 감소되거나 종결되고, 특정한 구현예에서, 환자가 질환의 증상의 부분적 또는 완전한 개선을 나타낼 때까지 상대적으로 짧은 간격의 상대적으로 높은 투여량(예컨대, 용량 당 약 1 내지 400 mg/kg의 항체, 보다 일반적으로 5 내지 25 mg의 투여량이 방사면역접합을 위해 사용되고 세포독성-약물 접합 분자용으로는 더 높은 투여량을 가짐)이 때때로 요구된다. 그런 다음, 환자는 예방적 요법을 투여받을 수 있다.
일 구현예에서, 대상체는 (예컨대, 벡터 내에) 본원에 개시된 폴리펩티드를 암호화하는 핵산 분자로 치료받을 수 있다. 폴리펩티드를 암호화하는 핵산의 용량은 환자 당 약 10 ng 내지 1 g, 100 ng 내지 100 mg, 1 ug 내지 10 mg, 또는 30-300 ug DNA의 범위이다. 감염성 바이러스 벡터용 용량은 용량 당 10 내지 100 이상의 비리온으로 다양하다.
치료제는 예방 또는 치료적 처치를 위한 비경구, 국소, 정맥내, 경구, 피하, 동맥내, 두개 내, 복강내, 비강내, 또는 근육내 수단에 의해 투여될 수 있다. 근육 내 주사 또는 정맥 내 주입이 본원에 개시된 항체의 투여를 위해 사용될 수 있다. 일부 구현예에서, 치료적 항체 또는 이의 단편은 두개골 내로 직접적으로 주사된다. 일부 방법에서, 항체 또는 이의 단편이 서방성 조성물 또는 디바이스, 예를 들면 MedipadTM 디바이스로 투여된다.
본원에 개시된 제제는 선택적으로 (예컨대, 예방적 또는 치료적) 치료를 필요로 하는 장애 또는 병태를 치료하는 데 효과적인 다른 제제와 조합하여 투여될 수 있다. 추가적인 제제는 당해 분야에 알려진 제제이고, 특정 장애에 대해 일상적으로 투여된다.
본원에 개시된 90Y 표지된 항체의 유효한 단일 치료 투여량(즉, 치료적 유효량)은 약 5 및 약 75 mCi 사이이고, 일 구현예에서, 약 10 및 약 40 mCi 사이의 범위이다. 131I 표지된 항체의 유효한 단일 치료 비 골수 절제 투여량은 약 5 및 약 70 mCi 사이이고, 일 구현예에서, 약 5 및 약 40 mCi 사이의 범위이다. 131I 표지된 항체의 유효한 단일 치료 절제 투여량(즉, 자가유래 골수 이식이 요구될 수 있음)은 약 30 및 약 600 mCi 사이이고, 일 구현예에서, 약 50 내지 약 500 mCi 미만의 범위이다. 키메라 변형 항체와 함께, 존재하는 뮤라인 항체의 더 긴 순환 반감기로 인해, 요오드 131 표지된 키메라 항체의 유효 단일 치료 비 골수 절제 투여량은 약 5 내지 약 40 mCi, 사이이고, 일 구현예에서 약 30 mCi 미만의 범위이다. 예컨대, 111In 표지에 대한 영상화 범주는 통상적으로 약 5 mCi 미만이다.
131I 및 90Y로 상당한 임상적 경험이 얻어졌지만, 다른 방사성표지들도 당해 분야에 알려져 있고, 유사한 목적을 위해 사용될 수 있다. 여전히 다른 방사성동위원소들이 영상화를 위해 사용된다. 예를 들어, 본 발명의 범주 내에 맞는 추가적인 방사성동위원소는, 이에 제한되지는 않으나, 123I, 125I, 32P, 57Co, 64Cu, 67Cu, 77Br, 81Rb, 81Kr, 87Sr, 113In, 127Cs, 129Cs, 132I, 197Hg, 203Pb, 206Bi, 177Lu, 186Re, 212Pb, 212Bi, 47Sc, 105Rh, 109Pd, 153Sm, 188Re, 199Au, 225Ac, 211A 213Bi를 포함한다. 이를 고려하면, 알파, 감마 및 베타 방출자는 모두 본 발명에 맞는다. 또한, 본 개시의 관점에서, 당업자는 불필요한 실험 없이 어떠한 방사성핵종이 선택된 치료의 과정에 맞는지를 용이하게 결정할 수 있다는 것이 제시된다. 이를 위해, 임상 진단에서 이미 사용되고 있는 추가적인 방사성 핵종은 125I, 123I, 99Tc, 43K, 52Fe, 67Ga, 68Ga뿐만 아니라 111In을 포함한다. 항체는 또한 표적화된 면역요법(Peirersz et al. Immunol. Cell Biol. 65: 111, 1987)에서의 잠재적인 사용을 위해 다양한 방사성핵종으로 표지된다. 이러한 방사성핵종은 188Re 및 186Re뿐 아니라 더 적은 범위로 199Au 및 67Cu를 포함한다. 미국 특허 제5,460,785호는 이러한 방사성동위원소를 고려한 추가적인 데이터를 제공하고, 참조로서 본원에 포함된다.
앞서 논의한 바와 같이, 본원에 개시된 항체 또는 이의 단편은 포유동물 장애의 생체 내 치료를 위한 약학적 유효량으로 투여될 수 있다. 이를 고려하면, 개시된 항체 또는 이의 단편이 활성 제제의 안정성을 증진하고 투여를 촉진하도록 제형화될 것이라는 것이 이해될 것이다. 일정한 구현예에서, 본 발명에 따른 약학적 조성물은 약학적으로 허용가능하고, 비독성인 멸균 담체(예를 들면, 생리학적 염수, 비독성 완충액, 보존제 등)를 포함한다. 본 출원의 목적에서, 치료제에 접합되거나 접합되지 않은 본원에 개시된 항체의 약학적 유효량은 표적에 효과적인 결합을 달성하고, 혜택, 예컨대, 질환 또는 장애의 증상을 개선하거나 물질 또는 세포를 검출하는 것을 달성하기에 충분한 양을 의미하는 것으로 간주되어야 한다. 종양 세포의 경우에서, 폴리펩티드는, 일정한 구현예에서, 종양 또는 면역반응성 세포 상의 선택된 면역반응성 항원과 상호작용할 수 있을 것이고, 이러한 세포 사멸의 증가를 제공할 것이다. 물론, 본원에 개시된 약학적 조성물은 유효량의 약학적 폴리펩티드를 제공하기 위해 단일 또는 다수 용량으로 투여될 수 있다.
본 개시의 범주와 일치하여, 본원에 개시된 항체는 인간 또는 다른 동물에 앞서 언급된 치료방법에 따라 치료 또는 예방 효과를 생성하기에 충분한 양으로 투여될 수 있다. 본원에 개시된 폴리펩티드는 이러한 인간 또는 다른 동물에 본원에 개시된 항체와 통상적인 약학적으로 허용가능한 담체 또는 희석제를 공지된 기술에 따라 조합하여 제조된 통상적인 투여 형태로 투여될 수 있다. 당업자는 약학적으로 허용가능한 담체 또는 희석제의 형태 및 특성이 조합될 유효 성분의 양, 투여의 경로 및 다른 잘 알려진 변수들에 의해 좌우된다는 것을 인식할 것이다. 또한, 당업자는 본 발명에 따른 하나 이상의 종의 폴리펩티드를 포함하는 칵테일이 특히 효과적인 것으로 판명될 수 있다는 것을 이해할 것이다.
VI. PAI-1 관련 질환 또는 장애의 치료 방법
본원에 개시된 항-PAI-1 항체, 또는 이의 단편은 PAI-1 활성을 상쇄하는 데 유용하다. 따라서, 다른 양태에서, 본 발명은 본원에 개시된 하나 이상의 항-PAI-1 항체, 또는 이의 항원 결합 단편을 포함하는 약학적 조성물을 필요로 하는 대상체에 투여함으로써 PAI-1 관련질환 또는 장애를 치료하기 위한 방법을 제공한다.
치료할 수 있는 PAI-1 관련질환 또는 장애는, 제한 없이, 신장, 간 또는 폐 섬유증과 같은 병리생리적 상태 또는 복부 유착 형성의 예방을 포함한다.
복부 간 유착의 발생은 인간 질병의 주요한 원인이다. 유착의 합병증은 생명을 위협하는 장폐색과 같이 심각할 수 있지만, 여성에서의 만성 골반염 및 불임도 또한 복막 유착의 흔한 후유증이다. 대부분의 유착은 수술에 의해 유도되지만, 일부 경우에는 염증, 자궁내막증, 화학적 복막염, 방사선 치료, 외래 물질 반응, 및 지속적 외래 복막 투석에 의해서도 유발되는 것으로 나타났다. 복막 손상은 섬유소 탈편재화를 야기하는 국소 염증 반응을 유발한다. 조직 플라스미노겐 활성인자(tPA)의 감소 및 플라스미노겐 활성인자 저해제 PAI-1 및 PAI-2의 증가에 의해 유발되는 복막 섬유소 용해 활성의 외상 후 부전이 침적된 섬유소가 영구적인 유착으로 조직화되도록 하는 것으로 추정된다.
세프라필름(Seprafilm®)과 같은 현재 이용가능하고 효과적인 치료 옵션은 개방형 접근(개복술)에서만 사용할 수 있고 복강경에서는 사용할 수 없다는 한계를 가진다. 잠재적인 치료에 대한 조사가 진행중이다.
일정한 예시적인 구현예에서, 본원에 개시된 항체는 말기 신부전의 주요 원인인 만성 신장 질환뿐 아니라 신장 섬유증 및 관련된 급성 신장 손상을 치료하기 위해 발생될 수 있다.
당업자는 통상적인 실험에 의해 PAI-1 연관 질환 또는 장애를 치료하기 위한 목적에 대해 효과적이고 비독성인 양의 항체(또는 추가적인 치료제)가 어떤 것인지를 결정할 수 있을 것이다. 예를 들어, 폴리펩티드의 치료적 활성량은 질환 상태(예컨대, 단계 I 대 단계 IV), 대상체의 연령, 성별, 의학적 합병증(예컨대, 면역억제 병태 또는 질환) 및 체중 및 대상체에서 원하는 반응을 유발하는 항체의 능력과 같은 인자들에 따라 달라질 수 있다. 투여 계획은 최적 치료 반응을 제공하기 위해 조정될 수 있다. 예를 들어, 몇몇의 나누어진 투여량이 매일 투여될 수 있거나, 치료 상황의 급박함에 의해 지시되는 바에 비례하여 용량이 감소될 수 있다. 그러나, 일반적으로, 유효 투여량은 체중 1 킬로그램 당 하루에 약 0.05 내지 100 밀리그램, 일 구현예에서, 체중 1 킬로그램 당 하루에 약 0.5 내지 10 밀리그램의 범위 내일 것으로 예상된다.
본원에 개시된 다른 양태들 및 그들의 구현예들은 서로 조합될 수 있다. 또한, 위에 기술된 임의의 양태들 및 그들의 구현예들은 아래에 기술된 임의의 특정 양태들 및 구현예들과 조합될 수 있다.
본 발명을 더욱 예시하는 역할을 하는 일부 특정 양태들 및 구현예들이 아래에 제공된다:
특정 양태들 및
구현예들의
기술
청구항 1
(a) 중쇄 구조형성(framework) 영역 및 중쇄 가변 영역(중쇄 가변 영역은 서열 번호 34를 포함하는 중쇄 CDR1 영역, 서열 번호 33을 포함하는 중쇄 CDR2 영역, 및 서열 번호 32를 포함하는 중쇄 CDR3 영역을 포함함); 및
(b) 경쇄 구조형성 영역 및 경쇄 가변 영역(경쇄 가변 영역은 서열 번호 37을 포함하는 경쇄 CDR1 영역, 서열 번호 145를 포함하는 경쇄 CDR2 영역, 및 서열 번호 35를 포함하는 경쇄 CDR3 영역을 포함함)을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체.
청구항 2
(a) 중쇄 구조형성 영역 및 서열 번호 86을 포함하는 중쇄 가변 영역; 및
(b) 경쇄 구조형성 영역 및 서열 번호 93을 포함하는 경쇄 가변 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체.
청구항 3
(a) 제2항의 항체의 상기 중쇄 가변 영역과 적어도 95% 동일한 중쇄 가변 영역; 및/또는
(b) 제2항의 항체의 상기 경쇄 가변 영역과 적어도 95% 동일한 경쇄 가변 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체.
청구항 4
제1항의 항체와 본질적으로 동일한 에피토프에 결합하는 단리된 단일클론 항체.
청구항 5
(a) 중쇄 구조형성 영역 및 중쇄 가변 영역(중쇄 가변 영역은 서열 번호 34를 포함하는 중쇄 CDR1 영역, 서열 번호 33을 포함하는 중쇄 CDR2 영역 및 서열 번호 32를 포함하는 중쇄 CDR3 영역을 포함함); 및
(b) 경쇄 구조형성 영역 및 경쇄 가변 영역(경쇄 가변 영역은 서열 번호 37을 포함하는 경쇄 CDR1 영역, 서열 번호 36을 포함하는 경쇄 CDR2 영역, 및 서열 번호 35를 포함하는 경쇄 CDR3 영역을 포함함)을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체.
청구항 6
제5항에 있어서, 중쇄 가변 영역이 서열 번호 6을 포함하고, 상기 경쇄 가변 영역이 서열 번호 7을 포함하는 항체.
청구항 7
제5항의 항체와 본질적으로 동일한 에피토프에 결합하는 단리된 단일클론 항체.
청구항 8
(a)
서열 번호 82를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편 및 서열 번호 91을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편;
(b)
서열 번호 83을 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 92를 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편;
(c)
서열 번호 84를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 93을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편;
(d)
서열 번호 85를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 91을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편;
(e)
서열 번호 85를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 93을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편;
(f)
서열 번호 86을 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 94를 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편;
(g)
서열 번호 87을 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 95를 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편;
(h)
서열 번호 88을 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 96을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편;
(i)
서열 번호 89를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 97을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편;
(j)
서열 번호 90을 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 98을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편;
(l)
서열 번호 86을 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 95를 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편;
(m)
서열 번호 89를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 93을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편; 또는
(n)
서열 번호 89를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 95를 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편을 포함하는, 인간 PAI-1에 특이적으로 결합하는 인간화 단일클론 항체.
청구항 9
(a)
서열 번호 22를 포함하는 중쇄 CDR1 영역, 서열 번호 21을 포함하는 중쇄 CDR2 영역, 및 서열 번호 20을 포함하는 중쇄 CDR3 영역을 포함하는 중쇄 가변 영역; 및 서열 번호 25를 포함하는 경쇄 CDR1 영역, 서열 번호 24를 포함하는 경쇄 CDR2 영역, 및 서열 번호 23을 포함하는 경쇄 CDR3 영역을 포함하는 경쇄 가변 영역,
(b)
서열 번호 28을 포함하는 중쇄 CDR1 영역, 서열 번호 27을 포함하는 중쇄 CDR2 영역, 및 서열 번호 26을 포함하는 중쇄 CDR3 영역을 포함하는 중쇄 가변 영역; 및 서열 번호 31을 포함하는 경쇄 CDR1 영역, 서열 번호 30을 포함하는 경쇄 CDR2 영역, 및 서열 번호 29를 포함하는 경쇄 CDR3 영역을 포함하는 경쇄 가변 영역,
(c)
서열 번호 40을 포함하는 중쇄 CDR1 영역, 서열 번호 39를 포함하는 중쇄 CDR2 영역, 및 서열 번호 38을 포함하는 중쇄 CDR3 영역을 포함하는 중쇄 가변 영역; 및 서열 번호 43을 포함하는 경쇄 CDR1 영역, 서열 번호 42를 포함하는 경쇄 CDR2 영역, 및 서열 번호 41을 포함하는 경쇄 CDR3 영역을 포함하는 경쇄 가변 영역,
(d)
서열 번호 46을 포함하는 중쇄 CDR1 영역, 서열 번호 45를 포함하는 중쇄 CDR2 영역, 및 서열 번호 44를 포함하는 중쇄 CDR3 영역을 포함하는 중쇄 가변 영역; 및 서열 번호 49를 포함하는 경쇄 CDR1 영역, 서열 번호 48을 포함하는 경쇄 CDR2 영역, 및 서열 번호 47을 포함하는 경쇄 CDR3 영역을 포함하는 경쇄 가변 영역,
(e)
서열 번호 52를 포함하는 중쇄 CDR1 영역, 서열 번호 51을 포함하는 중쇄 CDR2 영역, 및 서열 번호 50을 포함하는 중쇄 CDR3 영역을 포함하는 중쇄 가변 영역; 및 서열 번호 55를 포함하는 경쇄 CDR1 영역, 서열 번호 54를 포함하는 경쇄 CDR2 영역, 및 서열 번호 53을 포함하는 경쇄 CDR3 영역을 포함하는 경쇄 가변 영역,
(f)
서열 번호 58을 포함하는 중쇄 CDR1 영역, 서열 번호 57을 포함하는 중쇄 CDR2 영역, 및 서열 번호 56을 포함하는 중쇄 CDR3 영역을 포함하는 중쇄 가변 영역; 및 서열 번호 61을 포함하는 경쇄 CDR1 영역, 서열 번호 60을 포함하는 경쇄 CDR2 영역, 및 서열 번호 59를 포함하는 경쇄 CDR3 영역을 포함하는 경쇄 가변 영역,
(g)
서열 번호 64를 포함하는 중쇄 CDR1 영역, 서열 번호 63을 포함하는 중쇄 CDR2 영역, 및 서열 번호 62를 포함하는 중쇄 CDR3 영역을 포함하는 중쇄 가변 영역; 및 서열 번호 67을 포함하는 경쇄 CDR1 영역, 서열 번호 66을 포함하는 경쇄 CDR2 영역, 및 서열 번호 65를 포함하는 경쇄 CDR3 영역을 포함하는 경쇄 가변 영역,
(h)
서열 번호 70을 포함하는 중쇄 CDR1 영역, 서열 번호 69를 포함하는 중쇄 CDR2 영역, 및 서열 번호 68을 포함하는 중쇄 CDR3 영역을 포함하는 중쇄 가변 영역; 및 서열 번호 73을 포함하는 경쇄 CDR1 영역, 서열 번호 72를 포함하는 경쇄 CDR2 영역, 및 서열 번호 71을 포함하는 경쇄 CDR3 영역을 포함하는 경쇄 가변 영역; 또는
(i)
서열 번호 76을 포함하는 중쇄 CDR1 영역, 서열 번호 75를 포함하는 중쇄 CDR2 영역, 및 서열 번호 74를 포함하는 중쇄 CDR3 영역을 포함하는 중쇄 가변 영역; 및 서열 번호 79를 포함하는 경쇄 CDR1 영역, 서열 번호 78을 포함하는 경쇄 CDR2 영역, 및 서열 번호 77을 포함하는 경쇄 CDR3 영역을 포함하는 경쇄 가변 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체.
청구항 10
제8항 또는 제9항의 인간화 단일클론 항체와 본질적으로 동일한 PAI-1 상의 에피토프에 결합하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체.
청구항 11
플리스민 생성을 회복하는 방법으로서, 필요로 하는 대상체에게 약학적 유효량의 PAI-1 항체를 경구로, 주사용 용액에 의해 비경구로, 흡입으로, 또는 국소로 투여하는 단계를 포함하는 방법.
청구항 12
제11항에 있어서, 방법이 증가된 수준의 섬유증 조직을 포함하는 병태를 치료하는 방법.
청구항 13
제12항에 있어서, 병태가 섬유증, 피부 섬유증, 전신성 경화증, 폐 섬유증, 특발성 폐 섬유증, 간질성 폐 질환, 만성 폐 질환, 간 섬유증, 신장 섬유증, 만성 신장 질환, 혈전증, 혈관 및 동맥 혈전증, 심부 정맥 혈전증, 말초 사지 허혈, 파종혈관내응고 혈전증, 혈전 용해를 동반하는 그리고 동반하지 않는 급성 허혈성 뇌졸중, 또는 스텐트 재협착인, 방법.
청구항 14
제11항, 제12항, 또는 제13항에 있어서, PAI-1 항체가 전술된 청구항 중 어느 한 항의 항체를 포함하는 방법.
청구항 15
환자에게 경구로, 주사용 용액에 의해 비경구로, 흡입으로, 또는 국소로 투여하는 단계를 포함하는, PAI-1의 증가된 수준 또는 PAI-1에 대한 증가된 감수성에 의해 유발되는 병태를 치료하기 위한 약제의 제조를 위한 약학적 유효량의 PAI-1 항체의 용도.
실시예
본 발명은 하기의 실시예에 의해 더 예시되며, 이는 추가의 제한으로 이해되어서는 안 된다. 서열 목록, 도면들 및 모든 참조들의 내용, 본 출원 전체에 걸쳐 인용된 특허들 및 공개된 특허 출원들은 명백하게 참조로서 본원에 포함된다.
또한, 본 발명에 따라, 당해 분야의 기술 내에서 채용된 통상적인 분자 생물학, 미생물학 및 재조합 DNA 기술들이 존재할 수 있다. 이러한 기술들은 문헌에서 완전히 설명된다. 예컨대, 문헌(Sambrook, Fritsch & Maniatis, Molecular Cloning: A Laboratory Manual, Second Edition (1989) Cold Spring Harbor Laboratory Press, Cold Spring Harbor, New York (herein "Sambrook et al., 1989"); DNA Cloning: A Practical Approach, Volumes I and II (D.N. Glover ed. 1985); Oligonucleotide Synthesis (M.J. Gait ed. 1984); Nucleic Acid Hybridization [B.D. Hames & S.J.Higgins eds. (1985)]; Transcription And Translation [B.D. Hames & S.J. Higgins, eds. (1984)]; Animal Cell Culture [R.I. Freshney, ed. (1986)]; Immobilized 세포 And Enzymes [IRL Press, (1986)]; B. Perbal, A Practical Guide To Molecular Cloning (1984); F.M. Ausubel et al. (eds.), Current 프로토콜s in Molecular Biology, John Wiley & Sons, Inc. (1994))을 참조한다.
실시예
1:
하이브리도마
생성:
PAI
-1 단백질을 이용한 마우스의 면역화 및 항체 생성
인간(h) 및 시노몰구스(시노) 원숭이 활성 PAI-1(당화 및 비당화 형태)에 교차반응성일 수 있고, PAI-1의 저해 활성을 중화하고, 플라스민의 하류 생성을 회복함으로써 신장, 간 또는 폐 섬유증의 치료 또는 복부 유착 형성 및 켈로이드 흉터 형성의 예방에 효과적인 치료제가 될 수 있는 항체를 개발하였다. 단일클론 항체에 의한 PAI-1 저해 기능의 중화는 다음의 3가지 기전 중 하나에 포함되는 것으로 기술된다:(1) 입체 장애에 의한 tPA 또는 uPA에 대한 PAI-1의 차단, (2) PAI-1을 잠복성 형태로 전환, 또는 (3) PAI-1을 기질 형태로 전환.
a) 항원
PAI-1은 비트로넥틴에 대한 나노몰 이하의 친화도로의 결합에 의해 안정화된 활성 형태로 세포로부터 분비된다. PAI-1은 37℃에서는 수 분 내에 그리고 상온에서는 수 시간 내에 활성 형태로부터 잠복성 형태로의 자발적인 형태 변화를 거친다. 비트로넥틴에 결합하면, PAI-1은 형태 변화에 대해 더욱 저항성을 가지게 되는데, 이는 활성 형태에서 PAI-1의 반감기를 수 분에서부터 수 시간까지 연장한다. 면역화된 동물에서 활성 형태 PAI-1 반감기를 연장하고, 마우스 면역계가 활성 PAI-1 형태를 인식하도록 하기 위해, 비트로넥틴 및 PAI-1의 복합체를 면역화에 사용하였다.
곤충 세포에서 생성된 인간 당화 PAI-1를 Innovative Research(Cat# IGLYHPAI-A)로부터 구입하였다. 비트로넥틴(Cat# IHVN) 및 tPA(Cat# HTPA-TC)도 또한 Innovative Research로부터 구입하였다. 면역원을 생성하기 위해, PAI-1을 비트로넥틴과 1:1 몰 비로 1시간 동안 상온에서 항온배양하거나, tPA와 1:1 몰 비로 15분간 37oC에서 항온배양하였다. 모든 면역원은 멸균 염수를 희석제로 사용하여 제조하였다.
b) 면역화
당해 분야에 알려진 표준 하이브리도마 생성 프로토콜을 구현하여 항체를 생성하였다. 이전에 문헌에 기술된 표준 접근법은 PAI-1 단독 또는 PAI-1/tPA 복합체를 사용하였다. 본 발명자는 대신에 PAI-1의 활성 형태에 대한 항체를 생성하였다. PAI-1/비트로넥틴 복합체를 PAI-1에 대한 항체를 생성하기 위한 신규한 접근법으로써 사용하였다. 아래에 요약된 3가지 전략을 항체를 생성하기 위해 선택하였다:
(1) 하이브리도마를 생성하기 위한 융합 파트너로서 마우스 골수 세포주와 융합하기 위한 마우스 비장 세포를 얻기 위한 PAI-1/비트로넥틴 복합체로의 마우스의 고전적인 면역화;
(2) 하이브리도마를 생성하기 위한 융합 파트너로서 마우스 골수 세포주와 융합하기 위한 마우스 비장 세포를 얻기 위한 PAI-1/tPA 복합체를 이용한 마우스의 고전적인 면역화; 및
(3) 하이브리도마를 생성하기 위한 융합 파트너로서 마우스 골수 세포주와 융합하기 위한 마우스 비장 세포를 얻기 위한 PAI-1만을 이용한 마우스의 고전적인 면역화.
항원(PAI-1 단독, Vn/PAI-1 복합체, tPA/PAI-1 복합체) 당 3마리의 마우스를 본 연구에 사용하였다. 마우스는 9 내지 20주령 미처리(naive) 암컷 BALB/c 마우스(Charles River, 종 코드 028)였다. 0일에, 9마리의 마우스를 인산 완충 염수(phosphate-buffered saline, PBS) 중 PAI-1 단독, Vn/PAI-1 또는 tPA/PAI-1 복합체로 복강 내로 면역화하였다. 마우스 당 200 μl의 총 부피로 마우스 당 총 10 ug의 항원을 시그마 어쥬번트 시스템(Sigma Adjuvant System, Sigma cat #6322)과 1:1 부피 대 부피 비로 혼합하였다. 14일에, 마우스를 동일한 양의 항원으로 부스팅하였고, 0일째와 동일한 방식으로 준비시켰다. 21일에, PAI-1 특이적 항체 역가 평가를 위해 혈액 시료를 수집하였다. PAI-1/tPA 복합체로 면역화된 마우스는 PAI-1에 대하여 매우 낮은 특이적 반응성 및 높은 항-tPA 역가를 보였고, 아래의 융합에 사용되지 않았다.
51일에, 마우스 및 랫트 PAI-1 오소로그에 대해 가장 높은 역가를 가지면서, 가장 높은 항-PAI-1 특이적 항체 역가 및 PAI-1가 (즉, Vn 또는 tPA 중 하나)에 복합체화된 단백질에 가장 낮은 역가를 가지는 마우스를 융합을 위해 선택하였다. 융합을 위해 선택된 마우스를 전술된 바와 같이 PBS 중 PAI-1/ Vn 복합체 또는 PAI-1 단독(전술된 바와 같이 마우스 당 200 μl의 총 부피로 시그마 어쥬번트 시스템(Sigma cat #6322)과 1:1로 혼합된 마우스 당 총 10 ug의 항원)으로 부스팅하였다. 55일에 마우스를 CO2 챔버에서 희생시켰고, 심장 천자를 통해 혈액을 수집하였고, 하이브리도마 생성을 위해 비장을 수확하였다. 다른 4마리의 마우스는 더 늦은 시점(첫번째 마우스가 융합을 위해 사용된 후 2 내지 4달)에 동일한 절차를 거쳤다.
실시예 2에 기술된 ELISA 프로토콜(결합 ELISA)을 사용하여 PAI-1 단독 및 PAI-1/tPA의 경우 3마리의 마우스에서 그리고 PAI-1/Vn의 경우 2마리의 마우스에서 혈청 적정을 수행하였다.
PAI-1/tPA 복합체로 면역화된 마우스는 높은 특이적 역가 범주에 도달하지 않았고, 융합에 사용되지 않았다(표 3). 표 3에 나타낸 혈청 역가에 기반하여, PAI-1에 대해 높은 특이적 역가를 가지는 총 5마리의 마우스를 융합을 위해 선택하였다.
b) 융합
PAI-1에 대하여 가장 높은 특이적 역가를 가지는 다섯 마리의 마우스를 융합을 위해 선택하였다. 융합 날, 마우스를 CO2 챔버 내에서 희생시켰고, 심장 천자를 통해 혈액을 수집하였고, 비장을 제거하였고, 10 ml의 무혈청 하이브리도마 융합 배지(Iscove's Modified Dulbecco's Medium, IMDM 500 ml(Hyclone SH30259.01)를 함유하는 페트리 접시에 넣었다. 겸자로 탄력섬유 외피 밖으로 비장 세포를 짜내고, (초기의 회전을 포함하여) 10 ml의 무혈청 IMDM에서 2회 세척하였다.
세포를 Countess Automated Cell Counter로 계수하였다. 융합 파트너 세포(골수: FO(ATCC ref CRL-1646)) 및 비장 세포를, 그런 다음, 하나의 50 ml 튜브에 1:2 내지 1:10(세포 수에 의함)의 비율로 조합하여, 970 rpm(느린 회전)으로 10분간 회전시켜 느슨한 펠렛을 형성하였다. (37℃에서) 사전가열된 1 ml PEG(75 mM 헤피스(Hepes) 중 PEG 1500 50% w/v, Roche cat # 783641(10783641001))를 세포 펠렛에 1분의 기간 동안 적가하였고, 모든 PEG 방울이 첨가된 후 세포를 혼합하였다. 펠렛을 PEG와 함께 추가 1분 간 항온배양한 후, 10 중 첫번째 1 ml이 30초간 첨가되도록 10 ml의 무혈청 IMDM 배지를 1분 간 첨가하였다. 생존능을 보존하기 위해 세포를 970 rpm에서 10분 간 천천히 회전시켰다. 융합된 세포를 96-웰 플레이트 내 선택 배지(200 ml Gibco hybrioma(SFM # 12045), 20 ml 10% Hyclone SuperLow IgG Defined FBS(# SH30898.03), 2 ml 페니실린/스트렙토마이신, 4 ml(하이브리도마 융합 및 클로닝 보충제(Roche Diagnostics 11 363 735 001(50X)) 및 4 ml의 하이포잔틴-아미노프테린-티미딘(HAT)(Sigma-Aldrich # HO262(50X)))에 200 ul로 평판배양하였다. 융합체는 약 10 내지 14일 후 또는 웰 내 배지가 황색으로 변할 때 스크리닝에 대한 준비가 된 것이었다. 그런 다음, 발달된 하이브리도마로부터의 상층액을 PAI-1 및 PAI-1/Vn 복합체에 결합하는 항체의 존재에 대해 ELISA(실시예 2)로 시험하였다.
실시예
2:
PAI
-1 -
비트로넥틴
복합체에 대한 특이성에 대한
하이브리도마
상층액 스크리닝을 위한 결합 ELISA
선택된 다섯 마리의 마우스의 비장으로부터의 각각의 융합은, 일단계 일차 선별로서 PAI-1/Vn 복합체에 대한 결합에 대하여 스크리닝되어야 할 필요가 있는 약 5000개의 클론을 초래하였다. 비트로넥틴에 복합체화된 PAI-1에 특이적으로 결합하는 하이브리도마를 선택하기 위해, 하이브리도마 상층액의 일차 스크리닝은 PAI-1 또는 PAI-1-비트로넥틴 복합체에 대한 ELISA를 동시에 사용하여 수행하였다. ELISA를 위해 사용한 물질은 다음과 같았다: Immulon 4 HBX ELISA 플레이트(Dynax cat # N0541216); 인간 단량체 비트로넥틴 5 ug/ml(Innovative Research cat# IHVN); 당화 인간 PAI-1(활성 형태)(Molecular Innovations cat# GLYHPAI-A); 일부 융합 내 비당화 마우스 PAi-1(Molecular Innovations cat# MPAI-A); HRP-염소 항-마우스 IgG(H+L)였던 이차 항체(Jackson ImmunoResearch Labs # 115-035-166); 및, ABTS 기질: Roche Diagnostics(# 11 204 521 001).
사용된 대조군 항체는 다음과 같았다:
a) 33B8, PAI-1에 대한 마우스 단일클론 저해 항체(IgG1; Innovative Research cat# IMA-33B8);
b) 33H1, PAI-1에 대한 마우스 단일클론 저해 항체(IgG1; Innovative Research cat# IMA-33H1);
c) 31C9, PAI-1에 대한 마우스 단일클론 비저해 항체(IgG1; Innovative Research cat# IMA-31C9); 및
d) 1B7.11, IgG1 이소형 대조군 항체(항-TNP mAb -ATCC(Cat# TIB-191)로부터 구입한 하이브리도마 세포주로부터 직접 생산함).
ELISA 방법은 다음과 같았다: 플레이트를 PBS 중 5 ug/ml Vn으로 4℃에서 50 ul/웰로 밤새 코팅하였다; 다음날 플레이트를 PBS 중 1% 소 혈청 알부민(BSA/PBS) 200 ul으로 1시간 동안 차단하였다; 플레이트를 200 ul/웰 PBS로 4회 세척하였다; 1% BSA/PBS 중 2 ug/ml 활성 PAI-1를 플레이트에 50ul/웰로 첨가하였고 1시간 동안 항온배양하였다; 플레이트를 200 ul/웰 PBS로 4회 세척하였다; 1% BSA/PBS 중 항체 희석액 또는 원래의 96-웰 플레이트로부터의 하이브리도마 상층액을 ELISA 플레이트에 50 ul/웰로 첨가하였다; 플레이트를 상온(RT)에서 1시간 항온배양하였다; 플레이트를 200 ul/웰 PBS로 4회 세척하였다; 1% BSA/PBS 중 HRP-항-마우스 IgG 50 ul 1:2000을 첨가하였고 상온에서 1시간 항온배양하였다; 플레이트를 200 ul/웰 PBS로 4회 세척하였다; ABTS 기질(5 ml에 용해된 한 알)이 50 ul/웰로 플레이트에 첨가된 다음, BioTek Synergy HT 기기에서 OD405를 이용하여 플레이트를 판독하였다. 결합 ELISA에서의 항체 역가에 대한 통상적인 표준 곡선이 도 2에 나타나 있다. 항체 31C9, 33B8 및 33H1은 양성 대조군 역할을 하였고 IgG1은 음성 대조군 역할을 하였다. 표 4는 생성된 약 5000 클론 중에서, 675개의 클론이 PAI-1 및 PAI-1/Vn에 대한 결합에 대해 양성이었다는 것을 보여준다. 이러한 클론들을, 그런 다음, PAI-1 친화도에 대하여 스크리닝하였다.
실시예
3: 친화도
랭킹에
의한
하이브리도마
상층액의
비아코어
스크리닝
낮은 해리 속도를 가지는 고친화도 항체의 추가적인 선택을 비아코어로 수행하였다. 비아코어 하이브리도마 상층액 스크리닝은 (1) 항-마우스 고정 항-PAI-1 항체를 이용한 역방향 스크리닝 또는 (2) 리간드로서 또는 고정화된 Vn에 대하여 유리 PAI-1를 이용하는 정방향 스크리닝 검정 중 하나에 의해 수행될 수 있다.
사용된 기기는 실시간 생물분자 상호작용 분석(BIA)용으로 설계된 비아코어 2000 또는 비아코어 3000(GE Healthcare)이었다. 사용된 센서 칩은 표면에 카복시메틸화 덱스트란 매트릭스를 가지는 CM5 칩(GE Healthcare)이었다. 각각의 센서 칩은 4개의 평행한 흐름 셀(Fc)을 가진다. 모든 흐름 셀은 칩 제조를 위한 제조자의 프로토콜에 따라 표준 아민 커플링을 통해 항-마우스 IgG Fc mAb에 커플링되었다.
비아코어 역방향 스크리닝 검정에서, ELISA 양성 하이브리도마 상층액이 선택되었고, 비아코어 칩 표면 상에 주입되기 전에 0.2 μm 필터를 통해 여과되었다. 각각의 하이브리도마 상층액을 흐름 셀 Fc2 내지 Fc4 중 하나의 흐름 셀에 주입하였으며, 하이브리도마 상층액 내 IgG는 항-마우스 IgG Fc mAb에 의해 칩 표면에 포획될 것이되, Fc1은 참조 셀로서 단독으로 남겨놓았다. 그런 다음, PBS 중 인간 PAI-1 단백질을 Fc1 내지 Fc4에 주입하였다. PBS 완충액을 또한 블랭크(blank)로서 칩 표면에 주입하였다. Fc1 및 블랭크 완충액 시행의 신호를 감한 후, PAI-1 단백질에 대한 상층액으로부터의 항체의 결합 친화도(KD)/해리 속도(kd)를 분석하였고 Scrubber 2 소프트웨어를 사용하여 순위화하였다.
비아코어 정방향 스크리닝 검정에서, 정제된 인간 비트로넥틴 단백질을 CM5 칩 흐름 셀 Fc1 내지 Fc4에 고정하였다. 인간 또는 시노 PAI-1을 모든 흐름 셀 상에서 포획하였다. 그런 다음, 여과된 선택된 하이브리도마 상층액을, 참조 흐름 셀로 보존된 Fc1을 제외하고, 흐름 셀 당 하나씩 포획된 PAI-1에 주입하였다. PBS 완충액도 또한 블랭크로서 칩 표면에 주입되었다. Fc1 및 블랭크 완충액 시행의 신호를 감한 후, 비트로넥틴에 포획된 PAI-1에 대한 하이브리도마 상층액 내 항체의 결합 친화도를 분석하였고 Scrubber 2 소프트웨어(버전 2.0a, 2005; BioLogic Software, BioLogic Software Rty Ltd., 116 Blamey Court, Campbell, ACT 2612 호주)를 사용하여 순위화하였다.
표 5는 융합 A, B, C, D 및 E로부터의 양성 및 음성 항체 클론의 선택을 보여주는 것이다. 스크리닝된 항체 클론의 수가 많기 때문에 모든 데이터를 나타내지는 않았다. 인간 및 시노 PAI-1 단백질에 대하여 우수한 결합 해리 속도가 입증된 항체 클론(kd < 10 -4 1/s)만을 기능적 발색 검정을 위해 선택하였다.
실시예
4: tPA와
PAI
-1의 상호작용을 차단하는 항체를 선택하기 위한
하이브리도마
상층액
스크리닝을 위한 기능적 ELISA
기능적 항체의 선택을 가능하게 하기 위해, PAI-1에 결합만 하는 항체와 tPA 저해제로서의 PAI-1의 기능을 차단하는 항체를 구별하는 것을 가능하게 하는 새로운 ELISA(기능적 ELISA)를 개발하였다.
하이브리도마 상층액을 신규한 기능적 ELISA에서 스크리닝하여 tPA-PAI-1 상호작용을 차단하는 능력을 가진 상이한 클론부터 하이브리도마 상층액을 식별하였다. 기능적 ELISA의 설계는 다음과 같다: (1) 항체가 PAI-1에 결합은 하지만 항체 결합이 PAI-1과 tPA 사이의 공유 결합의 형성을 차단하지 않는 경우, 항-tPA 항체는 PAI-1를 통하여 플레이트에 결합된 tPA에 결합할 것이고 양성 판독을 제공한다; (2) 항체가 PAI-1을 차단하고, 이에 따라 PAI-1 형태를 변형하거나 입체 장애에 의해 tPA 상호작용을 차단하는 경우, 항-tPA 항체는 플레이트에 결합할 수 없을 것이고, 판독은 음성(낮은 OD405)일 것이다. 동시에, 하이브리도마 상층액을 실시예 2에 기술된 ELISA에서 PAI-1에 대한 결합에 대해 시험하였다. 하이브리도마 상층액 내 항체의 양을 모르기 때문에, 대조군 판독값보다 낮은(즉, 이소형 대조군 판독 값 아래) 것을 관심 있는 항체를 식별한 것으로 간주하였다. 상층액 내 다양한 항체 농도로 인해, 일부 경우에서는 차단은 단지 부분적이었다.
스트렙트아비딘으로 코팅된 플레이트(NUNC # 436014)를 50 ul/ml의 1% BSA/PBS 중 2 ug/ml 비오틴-PAI-1(N-말단 비오틴 표지를 가지는 인간 PAI-1, 활성 분획; Molecular Innovations cat # NTBIOPAI-A)과 함께 RT에서 2시간 동안 항온배양하였다. 플레이트를 200 ul 1% BSA/PBS로 RT에서 1시간 동안 차단하였고 200 ul/웰 PBS로 4회 세척하였다. 정제된 항체 희석액 및 하이브리도마 상층액을 웰에 50 ul/웰로 첨가하였고 15분 간 항온배양하였다. 플레이트를 200 ul/웰 PBS로 4회 세척하였다. 1 ug/ml 2쇄 tPA(Innovative Research cat# HTPA-TC)를 50 ul/웰로 플레이트에 첨가하였고, 30분 간 RT에서 항온배양하였다. 플레이트를 200 ul/웰 PBS로 4회 세척하였다. 1:3000 희석의 항-tPA HRP 접합 항체(Life Span Technologies, cat#LS-C39721)를 플레이트에 첨가하고 45분 간 항온배양하였다. 플레이트를 200 ul/웰 PBS로 4회 세척하였다. ABTS 기질(5 ml 중 용해된 하나의 정제; Roche Diagnostics # 11 204 521 001)을 50 ul/웰로 플레이트에 첨가하였고 발색을 위한 시간을 허용하였다. 플레이트를 OD405를 이용하여 BioTek Synergy HT 기기에서 판독하였다. IgG 이소형 대조군보다 낮은 값을 가지는 OD는 PAI-1에 대한 tPA 결합의 차단을 나타낸다.
일부 경우, 기능적 ELISA를 비아코어 상층액 스크리닝 이전에 수행하였는데, 하이브리도마 개발에 더욱 중요한 선택 단계로서의 역할을 하였다. 양성 대조군으로서 33H1, 음성 대조군으로서 IgG1 및 식별된 양성 항체 클론으로서 A44를 가지는 대표적인 곡선이 도 3에 나타나 있다.
200종 이상의 상층액을 스크리닝하였다. 표 6은 양성 및 음성 하이브리도마 상층액의 선택을 보여준다. 융합 당 약 10종의 하이브리도마가 기능적 ELISA에서 tPA에 대한 결합으로부터 PAI-1을 차단하는 능력을 보이는 것으로 나타났다. 하이브리도마 상층액으로부터의 데이터에 기반하여, 시퀀싱 및 배지 규모 항체 생성을 위한 하이브리도마를 선택하였다. D4는 비당화 PAI-1에는 잘 결합하지 않았지만, 당화 PAI-1에 대한 비아코어 결합에 기반하여 정제 및 시퀀싱을 위해 선택되었다. 정제된 항체를 친화도 역학에 대해서는 비아코어에서, 그리고 효능에 대해서는 발색 및 세포 검정에서 상업적으로 이용가능한 항체와 비교하여 추가로 특성분석하였다.
실시예 5: 5 '-RACE(cDNA 말단의 빠른 증폭)에 의한 시퀀싱 및 마우스 항체 정제
일련의 융합으로부터 생성된 특이적인 표적에 대한 항체는 동일한 서열을 가질 수 있다. 항체 생성의 초기 단계에 항체 유전자 시퀀싱을 수행함으로써, 임의의 가능한 중복 항체를 제거하였고 올바른 항체 유전자 서열이 항체 선택 및 인간화뿐 아니라 키메라 항체 형성을 유도하였다.
5'-RACE는 mRNA의 3' 또는 5' 말단의 미지 서열과 정의된 내부 자리 사이의 메신저 RNA 주형으로부터 핵산 서열을 증폭하기 위한 절차이다. 단측 특이성을 가지는 이러한 증폭 방법은 "한 방향" PCR 또는 "앵커(anchored)" PCR로 기술되어 있다. 리드 항체의 원래의 가변 뮤라인 항-인간 PAI-1 항체 서열을 5'-RACE cDNA 시퀀싱으로 결정하고 N-말단 단백질 시퀀싱으로 확인하였다.
가변 중쇄(VH) 및 경쇄(VL) IgG 서열을 결정하기 위해, 하이브리도마 세포로부터의 총 RNA를 제조자의 지시에 따라 RNeasy 미니 키트(QIAGEN, Cat No. 74104)를 사용하여 단리하였다. 간략하게, 세포(5 x 106 세포)를 350 ul의 키트의 RLT 완충액에 용해한 후 스핀 컬럼 상에서 총 RNA를 포획하였다. RNA를 키트의 TE 완충액에서 용리하고 얼음에 저장하였다.
제1 가닥 cDNA를, SMARTer™ RACE cDNA 증폭 키트(ClonTech, Cat No. 634923)를 사용하여 제조하였다. 5'-RACE 프로토콜을 제조자의 지시에 따라 수행하였다. VH 및 VL 사슬 cDNA를 SMARTer™ 키트에 의해 공급된 5'-프라이머와 아래에 열거된 3' VH 및 VL 유전자 특이적 프라이머를 이용하여 연쇄 중합효소 반응(polymerase chain reaction, PCR)으로 개별적으로 증폭하였다:
중쇄 3'- 프라이머: 5'-TATGCAAGGCTTACAACCACA -3'(서열 번호 105)
경쇄3'- 프라이머: 5'-CTCATTCCTGTTGAAGCTCTTGAG -3'(서열 번호 106)
증폭된 VH 및 VL 유전자를 TOPO TA 클로닝 키트(Invitrogen, Cat No. K4520-01)를 사용하여 TOPO 벡터에 개별적으로 클로닝하였다. 이러한 절차는 제조자의 지시에 따라 수행하였다. 박테리아를 형질전환하기 위해, 반응 혼합물을 수용성(competent) E. coli 세포에 첨가하고 얼음에서 20분 간 항온배양하였다. E. coli 세포 및 반응 혼합물을 함유한 튜브를 42℃에서 40초간 가열하였고, 250 마이크로리터의 리트(lit's) SOC 배지에 첨가하였다. E. coli를 37℃에서 300 rpm으로 진탕하면서 60분 간 항온배양한 후, 박테리아를 ml 당 100 마이크로그램의 암피실린을 함유하는 LB 아가 플레이트에 도말한 후 37℃에서 밤새 항온배양하였다.
PCR로 삽입된 VH 및 VL 유전자를 확인하여, 5종의 박테리아 클론을 선택하였고, 플라스미드 DNA 제조를 위해 ml 당 100 마이크로그램의 암피실린을 함유하는 LB 배지에서 증식시켰다. 플라스미드 DNA는 제조자의 지시에 따라 QIAprep 스핀 미니프렙 키트(QIAGEN, Cat No. 27104)를 사용하여 단리하였다. 하이브리도마의 VH 및 VL IgG 유전자를 생어(Sanger) 방법으로 시퀀싱하였고 CDR을 접촉 정의(MacCallum et al.)를 사용하여 결정하였다.
단일클론 항체를 CELLine 생물반응기 플라스크(Wilson Wolf Manufacturing Corp.; Cat. #CL350 또는 Cat # CL1000)에서 제조자의 지시에 따라 무혈청 배지(Gibco Cat. #12045)에서 생성하였고, 단백질 A/G 크로마토그래피(GE Healthcare Life Sciences, Cat. #28-4083-47 및 #28-4082-53)로 정제하였다. 정제된 항체를 상업적으로 이용가능한 항체와 비교하여 친화도 역학에 대해서는 비아코어로, 그리고 효능에 대해서는 발색 및 세포 검정으로 추가 특성분석하였다.
실시예
6: 정제된 항체를 이용한 기능적
발색
검정
정제된 항체를 PAI-1을 차단하는 능력에 대하여 발색 검정에서 시험하였다. PAI-1가 tPA 기능을 저해함으로, PAI-1를 차단하는 항체는 tPA 기능의 회복을 초래할 것이다. 발색 검정은 하나 이상의 펩티드 결합(들)을 가수분해함으로써 자연적인 기질(단백질 및 펩티드)에 작용하는 단백질 가수 분해 효소를 활용한다. 이러한 과정은 일정한 아미노산에 인접한 펩티드 결합만 절단된다는 점에서 대개 고도로 특이적이다. 발색 기질은 단백질 가수분해성 효소와 반응하여 정량가능한 색의 형성을 초래하는 펩티드이다. 발색 기질은 합성적으로 만들어지며, 효소에 대한 자연적인 기질과 유사한 선택성을 가지도록 설계된다. 발색 기질의 펩티드 부분에 부착된 것은 효소 절단 후 방출되면 색의 발생을 제공하는 화학적 기이다. 색 변화는 분광광도법으로 추적할 수 있고, 단백질 가수분해 활성에 비례한다.
tPA 저해제로서의 PAI-1 기능을 중화하는 항체의 능력을 확인하기 위해 발색 검정을 사용하였다. tPA는 pNA를 발색 기질 S2288로부터 방출시킬 수 있다. 용액 내 S228은 색을 나타내지 않지만, tPA에 노출되고 이어지는 pNA의 방출 후, 용액은 OD405에서 판독할 수 있는 황색을 발생시킨다. 색 형성은 효소 반응의 역학을 결정하기 위해 2~3 시간에 걸쳐 관찰될 수 있다. PAI-1은 농도 의존적 방식으로 tPA의 효소 활성을 차단할 수 있다.
2단계 발색 검정을 수행하였다. 시약이 기질 용액에 첨가되는 단계까지 모든 시약은 10x 농축이었다. 제1 단계에서, tPA 저해에서의 PAI-1 효능을 발색 검정(고정된 tPA 농도를 이용한 PAI-1 역가측정)을 사용하여 측정하였다. PAI-1 역가 곡선을 분석하여 tPA 활성을 차단하는 PAI-1에 대한 IC50을 결정하였다. 차후에, 곡선으로부터 계산된 IC80을 PAI-1 차단 기능을 중화하고 tPA 효소 활성을 회복하는 능력에 대한 추가적인 항체의 검증을 위해 선택하였다. 동일한 부피(25 ul)의 tPA(14 nM)(Innovative Research, Cat. No.IHTPA-TC) 및 당화(활성 형태) 인간 PAI-1(Molecular Innovations, Cat. No.GLYHPAI-A) 또는 비당화(활성 형태) 마우스 PAI-1(Molecular Innovations Cat. # IMPAI)를 조합하였고 고정된 농도의 tPA 및 108 nM에서부터 시작하는 PAI-1의 3배 계대 희석액을 사용하여 항온배양하였다. 모든 단백질 희석액은 1% BSA/PBS를 이용하여 제조하였다. 혼합물을 상온에서 96-웰 미세역가 플레이트의 웰에서 15분 간 항온배양하였다. 그런 다음, 제조자의 지시에 따라 희석된 200 ul 발색 기질 S2288(1.25 mM)(Chromogenix, Cat. No. S-820852)을 웰에 첨가하고, 405 nm에서 2시간 동안 매 10분마다 OD405 흡광도 변화를 기록하여 잔여 tPA 활성을 측정한다. 대조군의 경우, tPA의 부재(효소 반응 없음) 하에 배경을 측정하였고, 양성 대조군은 PAI-1가 없었고(100% tPA 활성), 음성 대조군은 tPA의 10배 초과의 PAI-1였다(tPA 활성의 완전한 차단). 33B8, A44, 33H1 및 IgG1에 대한 대표적인 곡선에 대해서 도 4를 참조한다.
제2 단계에서, 항체의 기능적 성질을 PAI-1 중화 검정을 사용하여 활성 PAI-1을 저해하고 tPA 기능을 회복하는 항체의 능력을 평가하여 결정하였다. 이러한 단계에서, 활성 PAI-1 12.5 ul(56 nM)를 동일한 부피의 1% BSA(Sigma, Cat. No. A3059)를 함유하는 PBS(Invitrogen, Cat. No.14190-144) 또는 2 uM부터 시작하는 항체의 3배 계대 희석액 중 하나와 함께 항온배양하였다. 대조군 및 미지 항체를 0.1 내지 300 nM 범위의 농도(5배 희석)에서 3 nM PAI-1과 함께 항온배양하였고 tPA를 혼합물에 첨가하였다. 모든 성분들을 10x 농축으로 상온에서 항온배양하였고, tPA에 의한 절단 시, 색을 투명한 색에서 황색으로 변화시키는 tPA 기질 S2288로 10배 더 희석하였다. 시료를 OD 405에서 37oC로 2시간 동안 매 10분마다 판독하였다. 혼합물을 96-웰 미세역가 플레이트의 웰 안에서 30분 간 상온에서 반응하도록 하여 항체-항원 복합체 형성을 달성하였다. 그런 다음, 25 ul의 tPA(tPA 활성의 IC80 저해에 상응하는 14 nM)를 웰에 첨가한 다음 상온에서 15분 간 항온배양하였다. 마지막으로, 제조자의 지시에 따라 희석된 200 ul 1.25 mM 기질 S2288을 믹스에 첨가하였다. 405 nm에서의 흡광도 변화를 2시간 동안 매 10분마다 기록하여 잔여 tPA 활성을 측정한다. 백 퍼센트 PAI-1 활성은 항체의 부재 하에 관찰되는 PAI-1 활성으로 정의된다. 항체에 의한 PAI-1 활성의 중화는 항체의 존재 하에 측정된 잔여 PAI-1 활성으로부터 계산된다. 대조군은 이소형 대조군(음성)으로서는 IgG1였고, 양성 대조군으로서는 33H1 mAb 및 33B8 mAb였다. B28, E16, E21, A75 및 IgG1에 대한 대표적인 곡선에 대해서는 도 5를 참조한다.
인간 tPA를 저해하는 인간 PAI-1의 오소로그를 2단계 발색 검정 시스템에서 시험하였다. 오소로그의 역가측정은 인간 PAI-1에 대해 전술된 바와 같이 수행하였고(역가측정의 대표적인 곡선에 대해서는 도 6을 참조) tPA 활성은 발색법으로 결정하였다(시노 및 마우스 PAI-1에 대한 33B8 및 A44의 대표적인 곡선에 대해서는 도 7을 참조). 검정에 사용된 인간 tPA의 최종 농도는 1.4 nM이었다. 12.5 ul 활성 PAI-1(56 nM)를 동일한 부피의 1% BSA를 함유하는 PBS 또는 2 uM에서부터 시작하는 항체의 3배 계대 희석액과 함께 항온배양하였다. 혼합물을 96-웰 미세역가 플레이트의 웰에서 30분 간 상온에서 반응하도록 하였다. 그런 다음, 25 ul의 tPA(14 nM)를 웰에 첨가하고 15분 간 상온에서 항온배양하였다. 반응을 종결시키기 위해, 200 ul tPA 기질 S2288(Chromogenix)(1.25 mM)을 혼합물에 첨가하였다. 오소로그 PAI-1은 Molecular Innovations으로부터 수득하였다: 마우스 PAI-1(야생형 활성 분획; cat# MPAI); 랫트 PAI-1(야생형 활성 분획; cat# RPAI); 및 토끼 PAI-1(안정적 돌연변이; cat# RbPAI-I91L) 시노 PAI-1(활성 시노 PAI-1)는 E. coli에서 직접 생산하였다. 비아코어 스크리닝에서의 토끼 및 랫트 오소로그의 불량한 해리 속도로 인해(데이터 미도시), 이러한 오소로그에 대한 항체의 스크리닝은 수행하지 않았다.
각각의 융합으로부터의 하나 이상의 항체는, 본 검정에서, 시노 및 인간 PAI-1 저해 기능을 차단하는 능력을 나타내었는데, 약 14종의 항체가 중간 내지 강한 차단 활성을 가졌다. A39 및 B28은 이러한 두 항체가 당화 hPAI-1은 차단하지만 인간 또는 시노 비당화 PAI-1에 대해서는 활성을 가지지 않는다는 점에서 독특한 프로파일을 가진다. C26을 제외하고 항체 중 어떤 것도 마우스 PAI-1 활성을 효율적(인간 PAI-1의 10배 이내)으로 차단할 수 없었다.
실시예
7: 단일클론 항체의 작용의 기전
단일클론 항체는 3가지의 상이한 기전으로 PAI-1을 저해할 수 있다: a) 입체 장애, b) 결합 시 PAI-1을 잠복성 형태로 전환함, 및 c) 저해제 대신 PAI-1을 tPA 형태에 대한 기질("기질 형태")로 전환함. PAI-1은 세린 프로테아제와의 상호작용 시 tPA와 공유 결합을 형성한다.
발색 검정 및 SDS-PAGE 기술을 항체 작용의 기전을 확인하기 위해 사용하였다. 단일클론 항체(또는 대조군 항체), PAI-1 및 tPA 사이의 반응은 기능적 발색 검정에 전술된 바와 같이 수행되었다. 시료를 라엠리(Laemmli) 시료 완충액과 혼합하고 비환원 조건 하에 SDS-PAGE 겔 상에 로딩하고 30분 간 시행하였다. 그 후, 겔을 쿠마시 블루(Coomassie blue)로 염색하여 단백질, 복합체 및 절단된 형태의 PAI-1을 시각화하였다. 알려진 작용 기전을 가지는 대조군 단일클론 항체를 비교기로서 사용하였다. 33B8는 PAI-1을 잠복성 형태로 전환하는 것으로 알려져 있고 33H1 PAI-1을 기질 형태로 전환하는 것으로 알려져 있다. 이러한 검정은 기질 형태를 양성으로 식별하지만, 잠복성 형태 또는 입체 장애와 구별할 수는 없다. 대표적인 SDS-겔이 도 8, 9 및 10에 나타나 있다.
A44, C26, C45 및 E21은 PAI-1 형태를 활성 형태에서 기질 형태로 전환함으로써 PAI-1 활성을 차단한다. A39 및 B109는 상이한 작용의 기전을 가지지만, 본 검정은 이들 항체가 PAI-1 활성을 PAI-1을 활성 형태에서 잠복성 형태로 전환하여 차단하였는지 또는 입체 장애에 의해 차단하였는지 여부를 구별할 수 없다.
실시예
8: 정제된 항체 결합 역학
역학 측정에서, 항체를 25℃에서 역으로 평가하였다. 본 역방향 검정에서는, PAI-1 항체가 CM5 칩 상에 제조된 항-마우스 IgG Fc 항체 표면에 포획된 후 PAI-1 단백질(인간 또는 시노)의 2x 계대 희석액을 40 nM에서부터 시작하여 주입하였다. 물질 이동 한계를 피하기 위해 50 ul/분의 빠른 유속을 선택하였다. 선택된 항체의 느린 해리 속도를 수용하기 위해 이천 초가 해리 시간으로 허용되었다. 이러한 칩은, 각 회차의 항체-PAI-1 결합 후, 글리신-HCl, pH 1.7 완충액으로 재생성되었다. 역학 데이터 분석은 비아코어 BIAevaluation 소프트웨어를 사용하여 수행하였다. 센서그램은 참조 흐름 셀 값 및 블랭크 완충액 값을 감함으로써 이중 참조되었다. 센서그램을 국소적인 Rmax를 가지는 시뮬레이션된 역학 1:1(Langmuir) 모델을 이용하여 적합하게 하였다. 시험한 항체에 대한 데이터는 아래의 표 9에 나타나 있다.
대표적인 항체의 결합 역학을 비트로넥틴과 PAI-1 복합체를 이용한 비아코어 정방향 검정을 통해 더 분석하고 비교하였다. 이러한 정방향 검정에서, 인간 비트로넥틴 단백질을 아민 커플링으로 흐름 셀 Fc1~Fc4 내 CM5 칩 상에 고정하였다. 인간 PAI-1을, 그런 다음, 리간드로서 흐름 셀 Fc2~Fc4 내 비트로넥틴 표면에 포획하였다. Fc1은 참조 셀로서 보존하였다. 항체를 40 nM에서부터 시작하여 2x로 희석하고 Fc1~4에 주입하였다. 역학 데이터 분석을 비아코어 BIAevaluation 소프트웨어를 사용하여 수행하였다. 센서그램을 우선 참조 셀 값 및 블랭크 완충액 값을 감함으로써 이중 참조하였고, 전반적인 Rmax를 이용하여 1:1(Langmuir) 모델로 적합하게 하여 사용하였다.
표 10의 데이터는 A44가 유리 인간 PAI-1뿐 아니라 비트로넥틴 복합체 내 PAI-1에도 결합한다는 것을 보여주었다.
실시예
9: 일차 인간 세포 내 기능적 검정
일차 인간 세포에 의한 하류 플라스민 생성을 회복하는 각각의 항체의 능력을 더 조사하기 위해, 플라스민 생성 검정을 사용하였다. 발색 검정에서 높은 효능 및 비아코어에서 양호한 친화도를 나타낸 항체만 본 검정에서 사용하여 시험하였다.
1일에, 인간 일차 간 성상 세포(Sciencell CA, cat no SC5300)를 20000 세포/웰로 기아(starvation) 배지(DMEM Gibco+글루타맥스-1 4.5 g/L D-글루코스, 피루베이트(31966-021), 0.2% 우태아 혈청 골드 PAA(A11-152))에 5% CO2하에 37℃에서 평판배양하였다. 2일에, PAI-1 활성을 중화하기 위해, 항체를 재조합 PAI-1(Molecular Innovation, cat# IGLYHPAI-A, 재조합 당화 인간 PAI-1, 최종 농도 5 nM)과 함께 상온에서 15분 간 사전항온배양하였다. 동시에, tPA(Molecular Innovations(cat# HTPA-TC), DMEM 중 5 nM, 레드 페놀을 포함하지 않음)를 37℃에서 15분 간 세포와 함께 항온배양하였다. 미결합 tPA를 세척한 후, PAI-1/mAb 혼합물을 세포에 첨가하였고, 그런 다음 잔여 tPA 활성을 글루-플라스미노겐/기질 혼합물(Glu-Pg: Sigma cat# 9001-91-6; 0.5 μM 최종 농도) 및 플라스민 발색 기질:(CBS00.65 Stago cat # 00128, 0.5 mM 최종 농도)을 첨가하여 측정하였다.
플라스민에 대한 플라스미노겐 활성화는 37℃로 온도 설정된 분광광도계(IEMS, Thermofisher)를 사용하여 A405/492 nm의 매 45초마다의 역학 판독으로 검출한다. Biolise 소프트웨어(Thermofischer)가 발색 기질 절단의 최대 속도: Vmax로 표시되는 플라스민 생성: 계산된 분 당 A405/492 nm(mDO/분)의 최대 속도를 계산한다. 그런 다음 PAI-1 저해를 참조로서 tPA 단독(100% 저해) 및 PAI-1(mAb 없음, 저해 없음)을 이용하여 계산하고, Biostat 스피드 소프트웨어를 사용하여 도표화하여 IC50 및 Imax를 계산한다.
실시예
10:
비아코어
경쟁 검정에 의한 항체 결합
에피토프
탐색
우수한 결합 및 차단 활성을 가지는 선택된 군의 항-PAI-1 항체를 비아코어 경쟁 검정에서 잠재적인 결합 에피토프에 대해 탐색하였다. 이러한 검정에서, 새로 식별된 항체뿐 아니라 인간 PAI-1에 대해 알려진 결합 자리를 가지는 몇몇 상업적으로 이용가능한 항-PAI-1 항체도 인간 PAI-1 단백질에 대한 결합에 대해 경쟁하도록 설정되었다. 각각의 항체를 표준 아미노 커플링 반응을 이용하여 비아코어 CM5 칩에서 흐름 셀에 고정하였다. 클론 B28을 제외한 모든 시험된 항체는 아민 커플링 후에도 결합 자리 활성을 보유하였다. 인간 PAI-1 단백질을 칩 상에 고정된 항체에 포획한 후, 분석물인 각각의 항체를 주입하였다. 고정화 항체와는 다른, 인간 PAI-1에 대한 결합 자리를 가지는 분석 항체만 비아코어에서 추가적인 결합 신호를 나타낼 것이다. 각각의 고정된 항체에 대한 경쟁 실험을 2회 반복하였고, 그 결과는 하기 표에 나타나 있다.
A44가 고정되고 PAI-1이 결합하는 경우, C45(분석 항체)는 A44에 의해 결합된 PAI-1에 결합할 수 없다. 따라서, C45는 PAI-1 상 A44가 결합한 동일한 결합 자리에 대해 경쟁(표 12에 "c/c"로 나타냄)하거나 PAI-1에 대한 A44 결합이 PAI-1에 대한 C45 결합을 방해한다. 이러한 분석은 실험이 반대의 순서로 반복될 때 확인된다. 구체적으로, C45가 고정된 항체이고 PAI-1에 결합하는 경우, 분석 항체로서 A44는 C45에 결합한 PAI-1에 결합할 수 없다(표 12에 "c/c"로 나타냄). 유사한 분석에서, A71 및 A75는 PAI-1 상의 동일한 자리에 대해 경쟁한다. 비아코어 분석은 A44 및 C45뿐만 아니라 A71 및 A75가 PAI-1에 결합할 때 서로 경쟁하거나 서로 방해한다는 것을 확인하였다.
반대로, 상업적으로 이용가능한 항체인 33H1 및 33B8은 A44와 경쟁하지 않는다. A44가 고정된 항체이고 PAI-1에 결합하는 경우, 33H1 및 33B8 모두 A44에 결합한 PAI-1에 여전히 결합할 수 있다(표 12에 "b/b"로 나타냄). 이는 역방향 실험에서 확인된다. PAI-1이 고정된 33H1 또는 고정된 33H8에 결합하는 경우, A44는 PAI-1에 여전히 결합할 수 있다. 따라서, 상업적인 항체 33H1 및 33B8은 PAI-1에 대한 A44 결합과 경쟁하거나 이를 방해하지 않는다.
흥미롭게도, 일부 고정된 항체(즉, B109)는 분석 항체(즉, 33B8)를 포획된 PAI-1 단백질에 대한 결합으로부터 차단하였지만; 분석 항체에 고정된 항체의 위치를 변경(예를 들어, 칩 상의 쌍을 뒤집음)하는 경우, 항체 쌍이 PAI-1에 대한 결합을 위해 더 이상 서로 경쟁하지 않았다. 예를 들어, B109가 PAI-1에 결합된 고정된 항체였던 경우, 33B8은 PAI-1에 결합할 수 없었다. 그러나, 33B8이 PAI-1 결합한 고정된 항체였을 때, B109는 PAI-1에 결합할 수 있었다. 이러한 결과에 대한 하나의 가능한 설명은 (예를 들어, B109가 고정된 항체이고 33B8이 분석 항체인 경우) 고정된 항체가 PAI-1에 결합할 때, PAI-1이 제2 또는 분석 항체에 대해 비우호적인 형태로 전환될 수 있고, 분석 항체를 결합으로부터 차단한다는 것이다. 그러나, 항체 쌍이 역전되면, 고정 항체는 PAI-1 형태가 거의 변하지 않는 방식으로 결합할 수 있고, 이에 따라 분석 항체가 결합된 PAI-1에 결합하는 것을 허용한다(즉, 분석 항체 B109는 고정 항체 33B8이 결합된 PAI-1에 결합할 수 있다). 따라서, 33B8과 B109 사이에서 관찰되는 경쟁은 PAI-1 상의 중첩되는 결합 자리로 인한 것이 아니라 B109에 결합할 때의 PAI-1에서 형태적인 변화에 기인할 것일 가능성이 있다.
다른 흥미로운 관찰은 B28은 아민 커플링에 의해 고정화되면 인간 PAI-1에 대한 결합을 소실한다는 것이었는데, 이는 B28의 CDR 영역이 일차 아민기(들)를 가지는 아미노산을 포함한다는 것을 시사한다.
실시예
11: 인간화를 위한 마우스 단일클론 항체의 선택
표 13은 수행된 5종의 융합으로부터의 가장 활성인 단일클론 항체를 특성분석한 시험관 내 데이터의 요약을 보여주는 것이다. 이러한 데이터에 기반하여, A44가 인간화를 위하여 선택되었는데, 이는 A44가 비아코어에서 가장 높은 친화도를 가지면서 발색 검정 및 플라스민 생성에서 가장 강한 항체였기 때문이다.
표 1 내 중쇄 및 경쇄서열이 도 12에서 정렬되고 IMGT에 의해 정의된 CDR은 굵은 글씨로 강조되어 있다. 표 13에 제시된 시험관 내 데이터에 기반하여, A44가 인간화를 위해 선택되었다.
실시예
12: 항-
PAI
-
1 A44
Fab의
조작: 인간화, 안정화 및 원치 않는 서열 모티프의 돌연변이
아래에서 논의된 몇몇 접근법이 PAI-1에 대한 A44 뮤라인 항체의 서열 모티프를 인간화, 안정화 및 최적화를 위해 선택되었다.
1) 인간화
사용된 인간화 프로토콜은, 그 전체가 참조로서 본원에 포함된 PCT/US08/74381(US20110027266)에 기술되어 있다. 뮤라인 A44의 가변 경쇄(VL) 및 가변 중쇄(VH) 서열을 분자 작용 환경(molecular operating environment, MOE; v. 2010.10; Chemical Computing Group)에서 항-PAI-1 A44 경쇄(LC) 및 중쇄(HC)의 상동성 모델을 구축하기 위해 사용하였다. 하기의 주형을 사용하였다: 경쇄구조형성 - 1D5I(구조형성 영역 내 94% 동일성), 중쇄 구조형성 - 3KSO(구조형성 영역 내 96% 동일성), L1 - 1D5I(94% 동일성), L2 - 1D5I(94% 동일성), L3 - 1AXS(72% 동일성), H1 - 1IC7(82% 동일성), H2 - 1MBU(68% 동일성) 및 H3 - 2WDB(62% 동일성). H3 루프는 모델링하기가 특히 어려웠는데, 이는 Trp이 첫 번째 잔기이기 때문이다. 또한, 2WDB는 비록 짧은 루프이지만, 루프의 시작점에 Trp을 가지고 H3 루프의 말단과 동일한 Phe-Asp-Tyr 서열도 가진다. His-99 및 Glu-105(LC)의 측쇄가 재구축되었고, 이어서 MOE에서 구현되는 표준 절차를 사용하여 모델을 최소 에너지화하였다. 뮤라인 A44의 최소화 3D 상동성 모델의 분자 동역학(MD) 시뮬레이션을, 이어서, Generalized Born implicit 용매 내에서 1.1 나노초(ns)간 500 K 온도에서 단백질 골격에 대한 제약을 가지고 수행하였다. 10가지의 다양한 형태를 마지막 1 ns 동안 매 100 피코초(ps)마다 이러한 제1 MD 시행으로부터 추출하였다. 그런 다음, 이러한 다양한 형태들은 2.3 ns 동안 단백질 골격에 대한 제약이 없고 300 K 온도인 MD 시뮬레이션에 제출하였다. 10회의 MD 시행 각각에서, MD 궤적으로부터의 마지막 2,000회의 스냅샷(매 ps마다 1회)을 다음으로 사용하여 각각의 뮤라인 A44 아미노산에 대해, 참조 중간점 위치에 비교하여 이의 평균 제곱근 편차(rmsd)를 계산하였다. 주어진 아미노산의 10종의 별개의 MD 시행에 대한 평균 rmsd를 모든 A44 뮤라인 아미노산의 전체적인 평균 rmsd와 비교하여, 당업자는 MD 중 보여지는 아미노산이 T-세포 수용체와 상호작용하여 면역 반응의 활성화를 담당하는 것으로 간주되는 것처럼 충분히 유연한지를 결정한다. CDR 및 이의 바로 옆 5 Å 부근을 제외하고, 뮤라인 A44 항체 내 37개의 아미노산이 유연한 것으로 확인되었다.
그런 다음, 20 ns(10 x 2 ns) 동안, 62개의 가장 유연한 뮤라인 A44 아미노산의 운동을, 각각이 10 x 2 ns MD 시뮬레이션을 시행한 49개의 인간 생식계열 상동 모델의 상응하는 유연한 아미노산의 운동과 비교하였다. 49개의 인간 생식계열 모델을 7개의 가장 공통적인 인간 생식계열 경쇄(vk1, vk2, vk3, vk4, vlambda1, vlambda2, vlambda3) 및 7개의 가장 공통적인 인간 생식계열 중쇄(vh1a, vh1b, vh2, vh3, vh4, vh5, vh6)를 조직적으로 조합하여 구축하였다. vk1-vh2 인간 생식계열 항체는 뮤라인 A44 항체의 유연한 아미노산에 비교하여 그의 유연한 아미노산에 대해 0.58의 4D 유사도를 나타내었다; vk1-vh2 생식계열 항체를, 따라서, 유연성 아미노산에 초점을 ?추어 F151 항체를 인간화하기 위해 사용하였다. v람다3-vh4 인간 생식계열이 두 번째로 높은 4D 유사성인 0.57을 나타내었고 또한 A44 항체의 인간화에 대한 기반으로 사용하였다. 뮤라인 A44와 vk1-vh2 아미노산 사이의 쌍 별 아미노산 결합을 위해, 2개의 서열을, 2종의 상응하는 상동성 모델의 알파 탄소의 최적 3D 중첩에 기반하여 정렬하였다. 뮤라인 A44와 v람다3-vh4 사이의 쌍 별 아미노산 결합을 유사한 방식으로 수행하였다. 도 13은 뮤라인 A44 경쇄와 vk1 및 v람다3의 정렬을 나타낸 것이다. 도 14는 뮤라인 A44 중쇄와 vh2 및 vh4의 정렬을 나타낸 것이다.
2) 안정화
a) 지식 기반 접근법
낮은 발생 빈도의 경쇄 및 중쇄의 아미노산 대 CDR을 제외한 그들의 정준 서열이 가장 빈번하게 발견되는 아미노산 내로 돌연변이화되도록 제안되었다(컴Gth > 0.5 kcal/mol; (E. Monsellier, H. Bedouelle. J. Mol. Biol. 362, 2006, p. 580-593)). LC 및 HC에 대한 컨센서스 돌연변이의 제1 목록은 가장 가까운 인간 생식계열(vk1-vh1b)에서 발견되는 아미노산으로 제한되었다. CDR의 바로 인접부(5 암스트롱의 "베르니어" 구역(J. Mol. Biol. 224, 1992, p. 487-499))내에 제시된 변화는 고려에서 제외하였다. 이는 LC 내 2개의 안정화 돌연변이(표 15 참조) 및 HC 내 다섯 개의 안정화 돌연변이(표 16 참조)를 초래하였다. 항 PAI-1 A44 항체를 잠재적으로 안정시키기 위한 이들 돌연변이의 다른 기준도 고려하였다. 이러한 범주는 표면 소수성의 호의적인 변화 또는 돌연변이의 분자 역학 기반 예상 안정화였다. 또한, 문헌(E. Monsellier & H. Bedouelle, J. Mol. Biol., 362, 2006, p. 580-593; B.J. Steipe et al. J. Mol. Biol, 1994, 240, 188-192)에서 추가적인 안정화 돌연변이도 성공적인 것이고 보고되었고 고려하였지만(표 17 및 18), 어떠한 추가적인 돌연변이도 제안되지 않았다.
b) 3D 및 MD-기반 접근법
3D 및 MD 기반 접근법들은 이전부터 보고되어 왔다(Seco J., Luque F.J., Barril X., J. Med. Chem. 2009 Apr 23:52(8):2363-71; Malin Jonsson et al., J. Phys. Chem. B 2003, 107:5511-5518). 항체의 소수성 영역은 이성분 용매(물 중 20% 이소프로판올, 20 ns 생성 시물레이션)에서 Fab의 분자 동역학 시뮬레이션을 분석함으로써 실험적으로 확인되었다. 슈뢰딩거(Schrodinger) 마에스트로 소프트웨어(v. 8.5.207) 내에서 소수성 표면 맵을 사용한 추가적인 분석을 완료하였다. 이러한 2가지 방법으로 분석된 단백질 표면은 상당히 친수성이다. 비록 이러한 2개의 기술에서 표면 상에 어떠한 소수성 패치를 제공하는 잔기도 존재하지 않았고, 이에 따라 어떠한 항응고 돌연변이도 제안되지 않았다.
3) 이식(grafting)에 의한 인간화
이식(grafting) 기술을 사용한 인간화는 이전에 보고되어 있다(P. T. Jones, P.H. Dear, J. Foote, M.S. Neuberger, G. Winter, Nature 1986, 321:522-525). 인간화는 항-PAI1 A44 가변 도메인 경쇄 및 중쇄에 가장 가까운 2종의 인간 생식계열을 확인함으로써 시작되었다. 이는 조직적으로 열거된 모든 인간 생식계열(카파 및 람다 사슬에 대한 V & J 도메인; 중쇄에 대한 V, D 및 J 도메인의 모든 가능한 조합)에 대한 BLAST 검색을 수행함으로써 이루어졌다. BLAST 검색은 미국국립생물정보센터(National Center for Biotechnology Information, NCBI)에 의해 제공되는 서열 정보 회수 및 분석(sequence information retrieval and analysis, SIRA) 서비스에 링크된 인터넷 어플리케이션을 사용하여 수행하였다.
가장 가까운 인간 생식계열은 항-PAI1 A44 가변 도메인 경쇄 및 중쇄에, 각각, 70% 및 67% 서열 동일성을 가지는 것으로 확인되었다. 내부 VBASE 생식계열 서열을 사용하여, 경쇄는 VI-O18(대략 64% 동일성) 유전자위(loci)에 가까운 것으로 밝혀졌고 중쇄는 VH4 서브패밀리의 4-30(대략 69% 동일성) 유전자위에 가까웠다. mA44 경쇄(A44LC) 및 IGVK1-33-01_IGKJ4-01(IGVK1)에 대한 CDR 영역(카바트 기반) 및 베르니어 잔기는 이탤릭체로 나타나 있다. 문헌(J. Mol. Biol., 1992, 224, 487)에 정의된 베르니어 잔기에는 밑줄이 처져있다. 인간화 돌연변이(굵은 글씨)는, 위에서 정의된 CDR & 베르니어 구역 잔기(또한 뮤라인 내에서 밑줄 쳐져 있음)를 제외한, 2개의 정렬된 서열의 쌍별 비교를 수행하여 수득하였다. 이식 접근법에 의한 인간화의 일부분으로서 뮤라인 경쇄로부터의 T46L 및 Q69T 및 뮤라인 중쇄 내 M2V(베르니어 구역 잔기)를 우세하게 보존된 인간 생식계열 서열로 돌연변이화하였다(LC5a, HC5a). 다른 변이체에서, 이러한 3개의 베르니어 구역 잔기가 원래의 뮤라인 서열(LC5b, HC5b)에서 볼 수 있는 바와 같이 유지되었다.
mA44 - 경쇄(서열 번호 141)
D I K M TQSPSS MYASLGERVT ITCKASQDIN SYLS WL QQKP GKSPK TLIY R
ANRSVDGVPS RFS G S G S GQ D Y SLTISSLEY EDMGIYYCLQ YDEFPPT F GG
GTKLEIK
IGKV1-33-01_IGKJ4-01(서열 번호 107)
DIQMTQSPSS LSASVGDRVT ITCQASQDIS NYLNWYQQKP GKAPKLLIYD
ASNLETGVPS RFSGSGSGTD FTFTISSLQP EDIATYYCQQ YDNLPLTFGG
GTKVEIK
mA44 - 중쇄(서열 번호 140)
E M QLQESGPS LVKPSQTLSL TCSVTG DSMT NGYWNWIRKF
PGNKLE YMG Y ITYSGSTYYN PSLKGR I S I T R N T SKNQ Y YL
QLSSVTTEDT ATYYC AR WHY GSPYYFDY W G QGTTLTVSS
IGHV4-59-02_IGHD6-13-01_IGHJ4-02(서열 번호 108)
QVQLQESGPG LVKPSETLSL TCTVSGGSVS SYYWSWIRQP
PGKGLEWIGY IYYSGSTNYN PSLKSRVTIS VDTSKNQFSL
KLSSVTAADT AVYYCARGYS SSWYYFDYWG QGTLVTVSS
다음으로 가까운 인간 생식계열은 항-PAI1 A44 가변 도메인 경쇄 및 중쇄에, 각각, 59% 및 58% 서열 동일성을 가지는 것으로 확인되었다. 내부 VBASE 생식계열을 사용하여, 이러한 경쇄는 VκIII -L6(약 56% 동일성) 유전자위에 가까운 것으로 밝혀지고, 중쇄는 VH6 서브 패밀리의 6-01 유전자위에 가깝다. CDR 영역(카바트에 기반함) 및 베르니어 영역은 이탤릭체로 표시되어 있다. 베르니어 영역(문헌(J. Mol . Biol., 1992, 224, 487)에 정의된 바와 같음)은 밑줄 처져 있다. 위에서 정의되고 굵은 글씨로 나타낸 CDR & 베르니어 구역 잔기(또한 뮤라인 내에 밑줄 처져 있음)를 제외한, 2개의 정렬된 서열을 쌍별 비교를 수행하여 인간화 돌연변이를 얻었다.
mA44 - 경쇄(서열 번호 141)
D I K M TQSPSS MYASLGERVT ITCKASQDIN SYLS WL QQKP GKSPK TLIY R
ANRSVDGVPS RFS G S G S GQ D Y SLTISSLEY EDMGIYYCLQ YDEFPPT F GG
GTKLEIK
IGKV3-11-02_IGKJ4-01(서열 번호 143)
EIVLTQSPAT LSLSPGE랫트 LSCRASQSVS SYLAWYQQKP GQAPRLLIYD
ASNRATGIPA RFSGSGSGRD FTLTISSLEP EDFAVYYCQQ RSNWPLTFGG
GTKVEIK
mA44 - 중쇄(서열 번호 140)
E M QLQESGPS LVKPSQTLSL TCSVTG DSMT N..GYWNWIR
KFPGNKLE YM G YIT..YSGS TYYNPSLKGR I S I T R N T SKN
Q Y YLQLSSVT TEDTATYYC A R WHYGSPYYF DY W GQGTTLT VSS
IGHV6-1-02_IGHD6-13-01_IGHJ4-02
(서열 번호 144)
QVQLQQSGPG LVKPSQTLSL TCAISGDSVS SNSAAWNWIR
QSPSRGLEWL GRTYYRSKWY NDYAVSVKSR ITINPDTSKN
QFSLQLNSVT PEDTAVYYCA RGYSSSWYYF DYWGQGTLVT VSS
4) 원치않는 서열 모티프의 돌연변이
다음의 모티프의 서열을 고려하였다: Asp-Pro(산 불안정 결합), Asn-X-Ser/Thr(당화, X=Pro이 아닌 임의의 아미노산), Asp-Gly/Ser/Thr(유연한 영역 내 숙신이미드/이소-asp 형성), Asn-Gly/His/Ser/Ala/Cys(노출된 탈아미드화 자리), 및 Met(노출된 영역 내 산화). 뮤라인 항-PAI1 A44의 VL & VH 도메인은 다음의 2개의 잠재적인 당화 자리를 소유한다: LC 내 (CDR2 내) N52RS 및 HC 내 N72TS. 하나의 노출된 탈아미드화 자리가 HC(N31G)의 CDR1 내에 존재한다. 다음의 숙신이미드 형성의 3개의 잠재적인 자리를 원래의 뮤라인 서열에서 확인하였다: LC 내 (CDR2의 말단의) D56G 및 HC 내 (CDR1 내) D27S 및 D89T. LC 문제성 모티프인 N52RS 및 D56G는 CDR2 내에 존재한다. 이러한 돌연변이가 CDR 내에 발생하기 때문에, 이들은 아래의 2개의 제안된 조작된 서열(LC2 및 LC4)에서 돌연변이에 의해 다루어진다. N52는 Gln으로 보존적으로 돌연변이되었고, D56은 Glu으로 돌연변이되었다. HC 내에는 4개의 문제성 잔기가 존재하고 있다. 첫번째 2개는 CDR1내에 발생한다: 잠재적인 숙신이미드 형성 자리인 D27S 및 탈아미드화 자리 N31G. 2개의 추가적인 문제성 모티프도 또한 제3 구조형성 영역 내에 발생한다. CDR1에서, D27은 숙신이미드의 형성을 피하기 위해 E로 돌연변이된 반면, N31은 Q로 바뀌었다. N72 및 D89를, 각각, Q 및 E로 바꾸었다. 이러한 문제성 모티프를 조작된 서열인 아래에 기술된 HC2a 및 HC4에서 다루었다. HC2b 변이체는 N31G 탈아미드화 자리의 돌연변이만 함유한다.
서열 중 어떠한 것도 임의의 공지된 인간 B 또는 T 세포 에피토프를 함유하지 않는다는 것을 보증하기 위해, 얻어진 인간화 서열을 IEDB 데이터베이스(immuneepitope.com. 의 범세계 웹에 밝혀짐; version June 2009; Vita R, Zarebesk L., Greenbaum J.A., Emami H., Hoof I., Salimi N., Damle R., Sette A., Peters B. The immune epitope 데이터베이스 2.0 Nucleic Acids Res. 2010, Jan, 38(데이터베이스 issue):D854-62. Epub 2009. Nov 11)에 대한 서열 유사성(BLAST 검색에서 수득된 결과에 대한 컷오프로써 70%의 서열동일성이 사용되고 인간 종으로부터의 결과만 고려하였음)을 블라스트분석하였다. DeClerck 등(국제 특허 번호 WO 2002034776)은 PAI-1의 항체 결합 에피토프를 개시하였는데, 이 중 어떤 것도 본원에 개시된 에피토프에 대해 문제가 있지 않았다.
뮤라인 A44 LC의 경우, Kirschmann 등(The Journal of Immunology, 1995, 155, 5655-5662)으로부터 하나의 인간 에피토프가 존재한다. 뮤라인 A44 LC는 아래에 나타낸 14개의 아미노산 스트레치와 약 71% 동일성을 보유한다. 대상 서열은 질량 분광계로 분석하지 않은 부분적인 서열이었다. 이러한 펩티드에 대해 어떠한 결합 데이터도 보고되지 않는다. 이러한 에피토프는 제안된 모든 LC 변이체에서 볼 수 있었다. 유사한 조사를 HC에 대하여 수행하였을 때 어떠한 잠재적인 문제성 에피토프도 확인되지 않았다.
5) 항-
PAI1
가변 도메인의 원래 서열
CDR은 굵은 글씨로 강조하고 베르니어 영역(Foote & Winter, J. Mol. Biol., 1992, 224:487-499에 의해 정의된 바와 같음)에는 밑줄이 처져 있다.
경쇄(서열 번호 142)
1 DIKMTQSPSS MYASLGERVT ITCKASQDIN SYLS WLQQKP GKSPKTLIY R
51 ANRSVDGVPS RFSGSGSGQD YSLTISSLEY EDMGIYYCLQ YDEFPPT FGG
101 GTKLEIKRAD AAPTVSIF
생식계열 지수(Germinality index) = IGKV1-33-01_IGKJ4-01 [VI-O18]와 70%
중쇄(서열 번호 140)
1 EMQLQESGPS LVKPSQTLSL TCSVTG DSMT NGYWNWIRKF PGNKLEYMG Y
51 ITYSGSTYYN PSLKGRISIT RNTSKNQYYL QLSSVTTEDT ATYYCAR WHY
101 GSPYYFDY WG QGTTLTVSS
생식계열 지수 = IGHV4-59-02_IGHD6-137-01_IGHJ4-02 [VH4 4-30]와 67%
6) 조작된 서열
4D 인간화 및 이식 접근법을 가장 가까운 2개의 인간 생식계열 서열에 적용하였다.
a) 조작된
경쇄
서열
LC1a는 가장 가까운 생식계열 서열인 vk1을 사용한 4D 인간화 방법으로부터 유래된 7개의 돌연변이를 함유한다. LC1b는 두번째로 가까운 인간 생식계열 서열인 vl3에 대한 4D 인간화로부터 유래된 12개의 돌연변이를 가진다. LC2는 LC1a와 비교하여 CDR2 내에 2개의 추가적인 돌연변이를 함유한다. 이러한 돌연변이는 잠재적인 당화 자리(N52RS) 및 잠재적인 숙신이미드 형성의 자리(D56G)를 다룬다. LC3은 추가적인 2개의 안정화 돌연변이를 가지는 가장 가까운 생식계열 서열에 대한 4D 인간화로부터의 돌연변이를 함유한다. LC4는 인간화, 안정화 및 원치않는 모티프 돌연변이를 조합한다. CDR 및 베르니어 구역은 이탤릭체이고, 베르니어 잔기에는 밑줄이 처져 있고, 인간화 돌연변이는 굵은 글씨이고, 문제성 모티프에는 이중으로 줄을 긋고, 안정화 돌연변이는 소문자로 나타나 있다. 도 16 및 17은 돌연변이의 요약을 보여주고 있다.
LC1a(서열 번호 91):
1 D I K M TQSPSS LSASVGDRVT ITCKASQDIN SYLS WL QQKP GKSPK TLIY R
51 ANRSVDGVPS RFS G S G S GQ D Y SLTISSLQP EDLGIYYCLQ YDEFPPT F GG
101 GTKLEIK
IEDB 데이터베이스 내에서 확인된 서열 LC1a에 대한 추가적인 인간 에피토프는 없다. LC1a 생식계열 지수 = IGKV1-33-01_IGKJ4-01 [VκI-O18]와 76%.
LC1b(서열 번호 92):
1 D I K M TQSPSS VSVSPGQTVT ITCKASQDIN SYLS WL QQKP GQSPK TLIY R
51 ANRSVDGVPS RFS G S G S GQ D Y SLTISSLQA MDEGIYYCLQ YDEFPPT F GG
101 GTKLTIK
위의 섹션 4에 기술된 에피토프에 추가하여, K39PGQSPKTLI는 KPGQPPRLLI에 70% 서열 동일성을 가진다(Kirschmann et al. J. Immun., 1995, 155, 5655-5662). 이러한 펩티드는 시험된 모든 HLA-DR 대립형질에 대하여 100,000 nM을 초과하는 IC50을 가지는 것으로 보고되어 있다. LC1b 생식계열 지수 = IGKV1-33-01_IGKJ4-01 [VκI-O18]와 67%.
LC2(서열 번호 93):
1 D I K M TQSPSS LSASVGDRVT ITCKASQDIN SYLS WL QQKP GKSPK TLIY R
51 AQRSVEGVPS RFS G S G S GQ D Y SLTISSLQP EDLGIYYCLQ YDEFPPT F GG
101 GTKLEIK
IEDB 데이터베이스에서는 서열 LC2에 대한 어떠한 추가적인 인간 에피토프도 발견되지 않았다.
LC2 생식계열 지수 = IGKV1-33-01_IGKJ4-01 [VκI-O18]와 76%.
LC3(서열 번호 94):
1 D I K M TQSPSS LSASVGDRVT ITCKASQDIN SYLS WL QQKP GKSPK TLIY R
51 ANRSVDGVPS RFS G S G S GQ D Y SLTISSLQP EDLatYYCLQ YDEFPPT F GG
101 GTKLEIK
IEDB 데이터베이스에서는 서열 LC3에 대한 어떠한 추가적인 인간 에피토프도 발견되지 않았다. LC3 생식계열 지수 = IGKV1-33-01_IGKJ4-01 [VκI-O18]와 78%.
LC4(서열 번호 95):
1 D I K M TQSPSS LSASVGDRVT ITCKASQDIN SYLS WL QQKP GKSPK TLIY R
51 AQRSVEGVPS RFS G S G S GQ D Y SLTISSLQP EDLatYYCLQ YDEFPPT F GG
101 GTKLEIK
IEDB 데이터베이스에서는 서열 LC4에 대한 어떠한 추가적인 인간 에피토프도 발견되지 않았다. LC4 생식계열 지수 = IGKV1-33-01_IGKJ4-01 [VκI-O18]와 78%.
LC5a(서열 번호 96):
1 D I Q M TQSPSS LSASVGDRVT ITCKASQDIN SYLS WL QQKP GKAPK L LIY R
51 ANRSVDGVPS RFS G S G S G T D Y TFTISSLQP EDIATYYCLQ YDEFPPT F GG
101 GTKVEIK
위의 섹션 4에 기술된 에피토프에 더하여, A43PKLLIYRAN은 APKLLIYAASSL에 대하여 80% 서열 동일성을 가진다(Kirschmann et al. J. Immun., 1995, 155, 5655-5662). 분자량은 이러한 펩티드에서 결정되지 않았고, 어떠한 결합 데이터도 보고되지 않았다. LC5a 생식계열 지수 = IGKV1-33-01_IGKJ4-01 [VκI-O18]와 85%.
LC5b(서열 번호 97):
1 D I Q M TQSPSS LSASVGDRVT ITCKASQDIN SYLS WL QQKP GKAPK TLIY R
51 ANRSVDGVPS RFS G S G S GQ D Y TFTISSLQP EDIATYYCLQ YDEFPPT F GG
101 GTKVEIK
IEDB 데이터베이스에서는 서열 LC5b에 대한 어떠한 추가적인 인간 에피토프도 확인되지 않았다.
LC5b 생식계열 지수 = IGKV1-33-01_IGKJ4-01 [VκI-O18]와 83%.
LC5c(서열 번호 98):
1 E I V M TQSPAT LSLSPGE랫트 LSCKASQDIN SYLS WL QQKP GQAPR TLIY R
51 ANRSVDGIPA RFS G S G S GQ D Y TLTISSLEP EDFAVYYCLQ YDEFPPT F GG
101 GTKVEIK
위의 섹션 4에 기술된 에피토프에 추가하여, K39PGQAPRTLI은 KPGQPPRLLI에 대해 80% 서열 동일성을 가진다(Kirschmann et al. J. Immun., 1995, 155, 5655-5662). 이러한 펩티드는 시험된 모든 HLA-DR 대립형질에 대하여 100,000 nM을 초과하는 IC50을 가지는 것으로 보고되어 있다. LC5c 생식계열 지수 = IGKV3-11-02_IGKJ4-01 [VκIII-L6]와 79%. 개략적인 모든 경쇄 돌연변이가 도 15에 나타나 있다.
b) 조작된
중쇄
서열
HC1a는 가장 가까운 인간 생식계열 서열에 대한 4D 인간화 방법으로부터 유래된 8개의 돌연변이를 함유한다. HC1b는 두번째로 가까운 생식계열 서열에 대한 4D 인간화 방법으로부터 유래된 6개의 돌연변이를 함유한다. 원치않는 서열 모티프를 다루기 위해, HC2a는 HC1a와 비교하여 4개의 추가적인 돌연변이를 함유한다. HC2b는 단지 CDR1 내 탈아미드화 자리(N31G)만을 다룬다. HC3은 추가의 5개의 안정화 돌연변이를 가지는 HC1a로부터의 인간화 돌연변이를 함유한다. HC4는 HC1a로부터의 인간화 돌연변이, HC3으로부터의 안정화 돌연변이 및 HC2a로부터의 문제성 모티프를 다루는 돌연변이를 함유한다. CDR 및 베르니어 구역은 이탤릭체이고, 베르니어 잔기에는 밑줄이 처져 있고, 인간화 돌연변이는 굵은 글씨이고, 문제성 모티프에는 두 줄을 그어져 있고, 안정화 돌연변이는 소문자이다.
HC1a(서열 번호 82):
1 E M TLKESGPT LVKPTQTLSL TCSVTG DSMT NGYWNWIRKF PGKALE YMG Y
51 ITYSGSTYYN PSLKGR I S I T R N T SKNQ Y YL TLSSVTTVDT ATYYC AR WHY
101 GSPYYFDY W G QGTTLTVSS
IEDB 데이터베이스에서는 서열 HC1a에 대해 어떠한 인간 에피토프도 확인되지 않았다. HC1a 생식계열 지수 = IGHV4-31-03_IGHD6-25-01_IGHJ4-02와 68%.
HC1b(서열 번호 83):
1 E M QLQESGPG LVKPSETLSL TCSVTG DSMT NGYWNWIRKF PGKGLE YMG Y
51 ITYSGSTYYN PSLKGR I S I T R N T SKNQ Y YL KLSSVTTADT ATYYC AR WHY
101 GSPYYFDY W G QGTTLTVSS
IEDB 데이터베이스에서 서열 HC1b에 대한 어떠한 인간 에피토프도 확인되지 않았다. HC1b 생식계열 지수 = IGHV4-31-03_IGHD6-25-01_IGHJ4-02와 73%.
HC2a(서열 번호 84):
1 E M TLKESGPT LVKPTQTLSL TCSVTG E SMT Q GYWNWIRKF PGKALE YMG Y
51 ITYSGSTYYN PSLKGR I S I T RQT SKNQ Y YL TLSSVTTVET ATYYC AR WHY
101 GSPYYFDY W G QGTTLTVSS
IEDB 데이터베이스에서 서열 HC2a에 대한 어떠한 인간 에피토프도 확인되지 않았다. HC2a 생식계열 지수 = IGHV4-31-03_IGHD6-25-01_IGHJ4-02와 67%.
HC2b(서열 번호 85):
1 E M TLKESGPT LVKPTQTLSL TCSVTG DSMT QGYWNWIRKF PGKALE YMG Y
51 ITYSGSTYYN PSLKGR I S I T R N T SKNQ Y YL TLSSVTTVDT ATYYC AR WHY
101 GSPYYFDY W G QGTTLTVSS
IEDB 데이터베이스에서 서열 HC2b에 대한 어떠한 인간 에피토프도 확인되지 않았다. HC2b 생식계열 지수 = IGHV4-31-03_IGHD6-25-01_IGHJ4-02와 67%.
HC3(서열 번호 86):
1 q M TLKESGPT LVKPTQTLSL TCSVsG DSMT NGYWNWIRqF PGKALE YMG Y
51 ITYSGSTYYN PSLKGR I t I T R d T SKNQ Y YL TLSSVTTVDT ATYYC AR WHY
101 GSPYYFDY W G QGTTLTVSS
IEDB 데이터베이스에서 서열 HC3에 대한 어떠한 인간 에피토프도 확인되지 않았다. HC3 생식계열 지수 = IGHV4-31-03_IGHD6-25-01_IGHJ4-02와 72%.
HC4(서열 번호 87):
1 q M TLKESGPT LVKPTQTLSL TCSVsG E SMT Q GYWNWIRqF PGKALE YMG Y
51 ITYSGSTYYN PSLKGR I t I T R Q T SKNQ Y YL TLSSVTTV ET ATYYC AR WHY
101 GSPYYFDY W G QGTTLTVSS
IEDB 데이터베이스에서 서열 HC4에 대한 어떠한 인간 에피토프도 확인되지 않았다. HC4 생식계열 지수 = IGHV4-31-03_IGHD6-25-01_IGHJ4-02와 70%.
HC5a(서열 번호 88):
1 Q V QLQESGPG LVKPSETLSL TCTVS G DSMT NGYWNWIRQP PGKGLE YMG Y
51 ITYSGSTYYN PSLKSR I T I S R N T SKNQY SL KLSSVTAADT AVYYC AR WHY
101 GSPYYFDY W G QGTLVTVSS
IEDB 데이터베이스에서 서열 HC5a에 대한 어떠한 인간 에피토프도 확인되지 않았다. HC5a 생식계열 지수 = IGHV4-59-02_IGHD6-13-01_IGHJ4-02 [VH4 4-59]와 84%.
HC5b(서열 번호 89):
1 Q M QLQESGPG LVKPSETLSL TCTVS G DSMT NGYWNWIRQP PGKGLE YMG Y
51 ITYSGSTYYN PSLKSR I T I S R D T SKNQ Y SL KLSSVTAADT AVYYC AR WHY
101 GSPYYFDY W G QGTLVTVSS
IEDB 데이터베이스에서 서열 HC5b에 대한 어떠한 인간 에피토프도 확인되지 않았다. HC5b 생식계열 지수 = IGHV4-59-02_IGHD6-13-01_IGHJ4-02 [VH4 4-59]와 84%.
HC5c(서열 번호 90):
1 Q M QLQQSGPG LVKPSQTLSL TCAIS G DSMT NGYWNWIRQS PSRGLE YMG Y
51 ITYSGSTYYA VSVKSR I T I N R D T SKNQ Y SL QLSSVTPEDT AVYYC AR WHY
101 GSPYYFDY W G QGTLVTVSS
IEDB 데이터베이스에서 서열 HC5c에 대한 어떠한 인간 에피토프도 확인되지 않았다. HC5c 생식계열 지수 = IGHV6-1-02_IGHD6-13-01_IGHJ4-02 [VH6 6-01]와 78%.
모든 중쇄 돌연변이에 대한 개략도가 도 16에 나타나 있다.
c)
중쇄
및
경쇄
변이체
서열의 조합
이식의 경우, 3가지 버전의 경쇄(LC5a, LC5b, LC5c) 및 3가지 버전의 중쇄(HC5a, HC5b, HC5c)를 생성하였다. LC5a는 가장 가까운 인간 생식계열 서열에 대한 이식으로부터 유래된 16개의 돌연변이를 함유하고 뮤라인 CDR 및 대부분의 뮤라인 베르니어 구역 잔기를 보유한다. 2개의 뮤라인 베르니어 잔기인 T46 및 N69는 어떠한 인간 생식계열 서열에도 존재하지 않고 보존적으로 돌연변이되었다. LC5b는 가장 가까운 인간 생식계열 서열에 대한 이식으로부터 유래된 14개의 돌연변이를 함유하고, 뮤라인 CDR 및 모든 뮤라인 베르니어 구역 잔기를 보유한다. LC5c는 두번째로 가까운 인간 생식계열 서열에 대한 이식으로부터 유래한 22개의 돌연변이를 함유하고, 뮤라인 CDR 및 모든 뮤라인 베르니어 구역 잔기를 보유한다.
HC5a는 가장 가까운 인간 생식계열 서열에 대한 이식으로부터 유래된 20개의 돌연변이를 함유하고, M2V를 예외로 한, 뮤라인 CDR 및 대부분의 뮤라인 베르니어 구역 잔기를 보유한다. Met는 인간 생식계열 서열 내 이러한 위치에서 매우 낮은 경향으로 나타난다. HC5b는 가장 가까운 인간 생식계열 서열에 대한 이식으로부터 유래된 20개의 돌연변이를 함유하고 뮤라인 CDR 및 모든 뮤라인 베르니어 구역 잔기를 보유한다. HC5c는 두번째로 가까운 인간 생식계열 서열에 대한 이식으로부터 유래된 23개의 돌연변이를 함유하고 뮤라인 CDR 및 모든 뮤라인 베르니어 구역 잔기를 보유한다.
총 10개의 조합이 준비되었다(표 19에 요약됨):
● LC1a x HC1a(가장 가까운 생식계열 서열에 기반한 4D 인간화를 다루는 돌연변이)
● LC1b x HC1b(두번째 가까운 생식계열 서열에 기반한 4D 인간화를 다루는 돌연변이)
● LC2 x HC2a(4D 인간화 및 원치않는 서열을 다루는 돌연변이)
● LC2 x HC2b(4D 인간화 및 원치않는 서열을 다루는 돌연변이)
● LC1a x HC2b(4D 인간화 및 원치않는 서열을 다루는 돌연변이)
● LC3 x HC3(4D 인간화 및 안정화를 다루는 돌연변이)
● LC4 x HC4(4D 인간화, 원치않는 서열 및 안정화를 다루는 돌연변이)
● LC5a x HC5a(CDR 및 삽입된 3개의 보존적 베르니어 변형을 보유하는 이식에 의한 인간화를 다루는 돌연변이)
● LC5b x HC5b(CDR 및 베르니어 영역을 보유하는 이식에 의한 인간화를 다루는 돌연변이)
● LC5c x HC5c(CDR 및 베르니어 영역을 보유하는 이식에 의한 인간화를 다루는 돌연변이)
요약하면, 인간화 과정 중 10종의 변이체가 생성되었다. 이러한 변이체들을 발현시키고 아래에 기술한 몇 개의 시험관 내 검정에서 특성분석하였다.
7) 인간화
변이체의
특성분석
위의 실시예에 제시된 컴퓨터 내(in silico) 모델링에 기반하여, 10종의 변이체를 생성하였다(4D 인간화에 의한 변이체 1~8 및 CDR 이식에 의한 변이체 9~10; 변이체 3 및 10은 두번째로 가까운 생식계열에 대하여 생성됨). HEK293 발현을 위해 인간화 A44의 경쇄 및 중쇄 DNA의 가변 영역을 제조하였다. 상응하는 DNA를 pXL 플라스미드(New England Biolabs; HC의 경우 NheI/Eco47III, LC의 경우 NheI/BsiWI)에 클로닝한 후 단백질을 생성하였다. HEK 발현을 위해 인간화 서열을 코돈 최적화하였고, GeneArt(Life Technologies의 자회사)에 의해 유전자를 합성하였다. 얻어진 플라스미드를 FreeStyleTM293 발현 시스템(Invitrogen, Cat#K9000-01)에 공동형질감염시키고 일시적으로 발현시켰다. 변이체 3 및 10은 매우 불량하게 발현되었고, 더 추적하지 않았다. 모든 다른 변이체를 발현시키고 단백질 A 칼럼을 사용하여 정제하였다. 분석 겔은 변이체 5 및 7의 중쇄 및 변이체 6 및 9의 경쇄의 부분적인 당화(약 5~10%)를 보여주었다(데이터 미도시). 남은 8종의 변이체를 hPAI를 사용한 발색 검정 및 인간 당화 PAI를 사용한 인간 성상 세포 내 플라스민 생성 검정에서 시험하였다. 결과는 표 23에 나타나 있다.
변이체 6 및 9는 플라스민 생성 검정에서는 가장 좋은 효능을 보였지만 경쇄에서 부분적(5~10%) 당화를 가졌다. 이러한 결과에 기반하여, 새로운 변이체 11~14를 변이체 6 및 9로부터의 중쇄와 변이체 5 및 7로부터의 경쇄의 조합을 사용하여 생성하였다. 표 24는 생성된 모든 변이체를 요약한 것이다.
불량하게 발현된 변이체 3 및 10을 제외한, 모든 변이체를 비아코어에서 인간 및 시노 PAI-1 및 비트로넥틴-PAI-1 복합체에 대하여 시험하였다. 데이터는 표 26에 제시되어 있다.
비아코어 데이터는 인간화 변이체 사이에서 유의한 차이를 나타내지 않았다. 변이체 8을 제외한 모든 인간화 변이체는 허용가능한 범위 이내로 시노 PAI-1 및 인간 PAI-1 및 비트로넥틴에 복합체화된 PAI-1에 친화도를 나타냈다. 모 A44와 비교하면, 인간화는 항체 친화도를 변화시키지 않는 것으로 보인다.
비록 인간화 변이체의 친화도 및 효능이 발색 및 비아코어 검정에서 유의하게 차이나지는 않았지만, 일부 변이체의 경우 세포 검정에서 플라스민 생성을 회복하는 변이체의 능력이 모 마우스 항체보다 유의하게 낮았다(아래의 발색 검정 및 세포 검정의 비교를 요약한 표 27 참조). 인간화 변이체 11~14를 세포 검정에서 PAI-1를 차단하는 능력에 대하여 시험하였다.
변이체 11 내지 14는 플라스민 생성 검정에서 양호한 효능을 나타냈고 추가적인 시험관 내 검정에서 추가로 특성분석하였다.
8) 인간 간에서 인간화
변이체의
특성 분석
인간화 변이체 11~14의 추가적인 스크리닝을 인간 혈장 및 인간 섬유증 간 시료로부터 내인적으로 생성된 인간 PAI-1을 사용하여 수행하였다.
PAI-1 활성을 이러한 세르핀이 96 웰 플레이트에 고정된 유로키나제와 안정한 복합체를 형성하는 능력을 측정함으로써 평가하였다. 미결합 PAI-1을 세척한 후, uPA-PAI-1 복합체를 다중클론 항PAI-1 항체의 사용으로 검출하였다. 그런 다음, 결합된 다중클론 항-PAI-1 항체(시료 내 활성 PAI-1에 비례함)를 홀스래디쉬 페록시다제 접합 이차 항체(Molecular Innovation Cat. No. HPAIKT)를 사용하여 검출하였다. 다양한 농도의 A44 인간화 변이체를 인간 또는 시노몰구스 재조합 PAI-1(0.31 nM 최종 농도)와 함께 상온에서 15분 간 항온배양한 다음, 전술한 ELISA를 사용하여 uPA-PAI-1 복합체에 의한 기능적 활성 PAI-1에 대해 시험하였다. 시료를 인간 PAI-1 표준과 비교하였다. 높은 활성 PAI-1 수준을 가지는 고 BMI 환자로부터의 인간 혈장을 4배 희석하고 증가하는 양의 A44 인간화 변이체와 함께 항온배양하였다. 남은 활성 PAI-1 수준을 ELISA에 의한 uPA-PAI-1 복합체 검출을 사용하여 결정하였다. 시노 재조합 PAI-1 중화를 또한 플라스민 생성에 의해 시험하여 교차 반응성을 확인하였다.
인간 섬유증 간 시료(Biopredic International, Rennes, 프랑스에 의해 제공됨(간 대장 전이물의 수술적 절제로부터 얻음))을 다음과 같이 균질화시켰다: 칭량된 동결 간 시료를 세라믹 비드(Cat No 03961-1-003, Bertin Technology, 프랑스)를 함유하는 건조 튜브에서 Precellys 균질기(Bertin Technology, 프랑스; 4℃, 6800 rpm 에서 2x30초)를 사용하여 균질화 한 다음 1 ml/g의 용해 완충액(TBS 중 NaCl 1.5M - Tris 완충 용액 0.1M Tris + 0.15M NaCl pH7.4)을 사용하여 용해하였다. 4℃에서 5000g로 10분 간 원심분리한 후, 상층액 내 간 용해물을 수확하고 -80℃에서 동결하여 저장하였다. 표준 BCA 검정을 이용한 총 단백질 농도 및 활성 & 총 PAI-1 수준(Mol Innov Cat No HPAIKT & Cat No MPAIKT-TOT에 의해 제공된 UK-PAI 복합체 ELISA에 의해 결정됨)을 제조자의 지시에 따라 A450 nm에서의 표준 인간 PAI-1 농도를 Biostat Calibration 소프트웨어를 사용하여 도표화함으로써 수행하였다. 2.5 nM의 활성 PAI-1까지 희석된 간 용해물과 함께 항온배양된 증가하는 농도의 A44 인간화 변이체를 이전에 기술한 바와 같이 평가하였고 데이터를 분석하였다. PAI-1 활성의 저해(mAb가 없는 PAI-1 활성이 0% 저해가 됨, IgG1로의 유의하고 용량 의존적인 PAI-1의 저해는 발생하지 않음)를 각 mAb 농도에 대하여 계산하였다. PAI-1 활성의 저해 백분율을 mAb 농도의 함수로 도표화하였고 IC50을 Biostat speed 소프트웨어를 사용한 Imax로 결정하였다. 데이터는 도 17 및 표 29에 나타나 있다.
위의 데이터에 기반하여, A44-hv11이 추가적인 구조 연구 및 추가적인 시험관 내 및 생체 내 연구에서 더 특성분석하기 위해 선택되었다.
실시예
13: 이식에 의한
APG
항체의 인간화
이식 기술을 이용한 인간화는 이전에 보고되어 있다(P. T. Jones, et al., Nature 1986, 321:522-525). 항-PAI1 뮤라인 항체 APG의 인간화는 독일 특허 출원 번호 DE2000153251로부터의 뮤라인 경쇄(서열 번호 148) 및 뮤라인 중쇄(서열 번호 149)로 시작되었는데; 이러한 뮤라인 항체는 또한 문헌(Debrock et al., Biochimica et Biophysica Acta, 1337(2):257-266(1997))에도 기술되어 있다. 뮤라인 항체의 생식계열 및 표준적(canonical) 클래스의 HC 및 LC 사슬을 확인하여 muIGHV1-39 및 muIGKV14-11을 각각 산출하였다. 다음으로, 항-PAI1 APG 가변 도메인 경쇄 및 중쇄에 가까운 인간 생식계열의 목록을 확인하고 동일성 백분율로 순위화하였다. 두 단계 모두 조직적으로 열거된 전체 인간 생식계열(카파 및 람다 사슬에 대한 V & J 도메인; 중쇄에 대한 V, D 및 J 도메인의 모든 가능한 조합)에 대한 BLAST 검색을 수행함으로써 이루어졌다. BLAST 검색은 http://www.imgt.org.(Ehrenmann, et al. Cold Spring Harbor Protocols 2011.6(2011) 참조)에 제공된 IMGT/DomainGapAlign 도구를 사용하여 수행하였다. 가장 가까운 인간 생식계열은 항-PAI1 APG 가변 도메인 경쇄 및 중쇄에 각각 67.4% 및 63.3% 서열 동일성을 가지는 것으로 확인되었다. IMGT 데이터베이스를 사용하여, 경쇄는 HuIGKV1-33에 가까운 것으로 밝혀졌고 중쇄는 HuIGHV1-46에 가까웠다. 매칭되는 표준적 클래스를 가지는, 항-PAI1 APG 가변 도메인 중쇄에 가장 가까운 인간 생식계열은 62.2%의 서열 동일성을 가지는 HuIGHV7-4-1인 것으로 밝혀졌다.
모 뮤라인 APG(mAPG) 경쇄(서열 번호 148), IGKV1-33-01_IGKJ4-01(IGKV1a)(서열 번호 107) 및 IGKV1-33-01_IGKJ2-02(IGKV1b)(서열 번호 150)에 대한 CDR 영역(APG에 대한 카바트 및 IMGT의 조합에 기반함) 및 베르니어 잔기는 이탤릭체로 나타나 있다(아래의 표 30 참조). 문헌(Foote, et al. J. Mol . Biol. 224(2):487-99(1992))에 정의된 베르니어 잔기에는 밑줄이 처져 있다. 인간화 돌연변이(굵은 글씨)는 위에 정의된 CDR & 베르니어 구역 잔기(또한 mAPG 서열 내 밑줄 처짐, 표 30)를 제외한 2개의 정렬된 서열의 쌍별 비교를 수행함으로써 얻어졌다. 뮤라인 APG 항체에 대해 어떠한 추가적인 조작도 수행되지 않았다. 이러한 인간화 항체를 APGv2 및 APGv4로 명명하였다.
조작된 서열
4D 인간화 및 이식 접근법을 전술된 인간 생식계열 서열 매치에 적용하였다. 조작된 경쇄 서열의 경우, APGv2가 인간 IGKV1-33 생식계열(APGv2 생식계열 지수 = IGKV1-33-01_IGKJ2-01와 94%)에 이식된 뮤라인 경쇄 CDR을 함유한다. 조작된 중쇄 서열의 경우, APGv2 및 APGv4rk가 각각 인간 IGHV7-4-1 및 IGHV1-46 생식계열(APG_VH2 생식계열 지수 = IGHV7-4-1-02_IGHD6-25-01_IGHJ4-02와 91%; APG_VH4 생식계열 지수 = IGHV1-46-01_IGHD6-25-01_IGHJ4-02와 91%)에 이식된 뮤라인 중쇄 CDR을 함유한다. 위의 표 30을 참조한다.
중쇄
및
경쇄
변이체
서열의 조합
이식을 위해, 하나의 버전의 경쇄(APGv2_VL2; 서열 번호 153) 및 두개의 버전의 중쇄(APGv2_VH2; 서열 번호 154 및 APGv4_VH4; 서열 번호 155)를 생성하였다. APG_VL2는 가장 가까운 인간 생식계열 서열에 대한 이식으로부터 유래한 15개의 돌연변이를 함유하고 뮤라인 CDR 및 베르니어 구역 잔기를 보유한다. APG_VH2는 매칭되는 표준적 클래스를 가지는 가장 가까운 인간 생식계열 서열에 대한 이식으로부터의 21개의 돌연변이를 함유하고, 뮤라인 CDR 및 베르니어 구역 잔기를 보유한다. APG_VH4는 가장 가까운 인간 생식계열 서열에 대한 이식으로부터의 20개의 돌연변이를 함유하고, 뮤라인 CDR 및 베르니어 구역 잔기를 보유한다. 이러한 이식 프로토콜을 위한 CDR에 대한 제한은 문헌에서 이용가능한 다양한 상이한 정의에 엄밀하지 않게 기반한다.
●
APG_VL2 x APG_VH2(CDR 및 베르니어 영역을 보유하는 이식에 의한 인간화를 다루는 돌연변이)
●
APG_VL2 x APG_VH4(CDR 및 베르니어 영역을 보유하는 이식에 의한 인간화를 다루는 돌연변이)
이러한 인간화 캠페인 중 2종의 mAPG 변이체를 생성하였고, 이를 APGv2 및 APGv4로 명명하였다. 이러한 변이체를 발현시키고 아래에 기술된 몇몇 시험관 내 검정에서 특성분석하였다.
실시예
14: 표면
플라스몬
공명에 의한
APG
항체에 대한 친화도 역학
마우스 APG 및 2종의 인간화 변이체(APGv2 & APGv4)의 인간 당화 PAI-1(GLYHPAI-A, Molecular Innovation)에 대한 친화도를 비아코어 2000 기기(GE Healthcare, Uppsala, 스웨덴)를 사용한 표면 플라스몬 공명(SPR)으로 조사하였다.
우선, 센서 칩 CM5(GE Healthcare, Uppsala, 스웨덴)의 표면을 마우스 및 인간 항-Fc(항-인간 IgG(Fc) 항체 & 항-마우스 IgG 항체 키트, GE Healthcare)의 포획을 위해 일상적인 아미노 커플링을 사용하여 제조하였다. 모든 단일클론 항체(mAb)를 HBS-EP 러닝 완충액을 이용하여 5 nM까지 희석하였다. 각각의 정제된 mAb를 3분 간 상이한 흐름 셀 표면에서 포획하였다. 인간 PAI-1을, 중간에서는 짧은 해리 시간 그리고 마지막에는 긴 해리 시간을 가지고(접촉 시간: 120초, 짧은 해리: 90초; 긴 해리: 1800초, 유속: 50 μl/분) 다양한 농도(2.5, 5, 10, 20 및 40 nM)로 주입하였다. 각 회차의 항체-PAI-1 결합 후 글리신-HCl, pH 1.7 완충액으로 칩을 재생성하였다. 역학 데이터 분석을 비아코어 BIAevaluation 소프트웨어를 사용하여 수행하였다. 참조 흐름 셀 값 및 블랭크 완충액 값을 감함으로써 센서그램을 이중참조하였다. 센서그램을 국소 Rmax를 가지는 시물레이션된 역학 1:1(Langmuir) 모델을 사용하여 적합하게 하였다. (도 19 참조). 3종의 APG 항체에 대한 데이터는 표 31에 나타나 있다.
실시예
15: 인간 혈장에서
APG
항체의 특성분석
마우스 APG 및 인간화 변이체 APGv2 및 APGv4를 본원에 개시된 기능적 검정에 따라 PAI-1을 차단하는 능력에 대해 스크리닝하였다(예를 들어, 위의 실시예 6 및 9를 참조). 간략하게, PAI-1 활성을 이러한 세르핀이 96 웰 플레이트에 고정된 유로키나제와의 안정한 복합체를 형성하는 능력에 의해 평가하였다. 미결합 PAI-1을 세척한 후, uPA-PAI-1 복합체를 다중클론 항PAI-1 항체의 사용에 의해 검출하였다. 그런 다음, 결합된 다중클론 항-PAI-1 항체(시료 내 활성 PAI-1에 비례함)를 제조자의 지시에 따라 홀스래디쉬 페록시다제 접합 이차 항체(Molecular Innovation, Cat # HPAIKT)를 사용하여 검출하였다.
다양한 농도의 APG 인간화 변이체(APGv2, APGv4) 또는 모 마우스 APG 항체를 15분 간 상온에서 높은 활성 PAI-1 수준을 가지는 미희석 인간 혈장과 함께 항온배양하였다. 잔여 활성 PAI-1 수준을 제조자의 지시에 따라 전술된 ELISA(예를 들어, 실시예 6 참조)에 의한 uPA-PAI-1 복합체 검출을 사용하여 결정하였다.
PAI-1 활성의 저해를 각각의 mAb 농도에 대해 결정하였다. PAI-1 활성의 저해 백분율을 APG 인간화 변이체(APGv2, APGv4) 또는 모 마우스 APG 항체의 농도의 함수로서 도표화하였다. 3회의 독립적인 실험(이중) 후, Biostat 스피드 소프트웨어를 IC50 및 Imax를 결정하기 위해 사용하였다(도 20 참조). 데이터는 아래의 표 32에 나타나 있다.
실시예
16: 인간 혈장 내
응괴
용해 검정:
A44V11
,
mAPG
, 및
APG
변이체
활성
뇌졸중을 지닌 환자에서는 종종 섬유소용해성 시스템이 변형된다. 응괴 용해 검정은 섬유소 분해의 정도를 측정함으로써 섬유소용해성 활성을 결정하기 위해 사용될 수 있다. 일반적으로, 문헌(Lindgren, A. et al. Stroke 27:1066-1071(1996))을 참조한다. 응괴 용해 검정은 다른 곳에서도 자세히 기술되어 있다. 예를 들어, 문헌(Beebe, et al. Thromb . Res. 47:123-8(1987); Tilley et al., J. Vis . Exp.67:e3822)을 참조한다.
A44V11 및 기타 PAI-1 중화 항체의 기능적 활성을 인간 혈장 응괴 용해 검정을 사용하여 결정하였다. 간략하게, 본원에 적용된 검정은 tPA 및 응괴 용해를 저해하는 것으로 알려진 농도의 PAI-1의 존재 하에 조직 인자/Ca2+의 혼합물을 사용한 응괴 형성을 유도한다. 섬유소 중합은 340 nm에서의 흡광도 측정에 의해 검출되었던 비탁도의 증가를 유도한다. 응괴 용해를 회복하는 항체의 능력을, 증가하는 용량의 항체를 정상적인 인간 혈소판 부족 혈장과 함께 항온배양하여 결정하였다.
간략하게, 응괴 용해 실험을 미세역가 플레이트에서 수행하였다. 시트르산 인간 혈장(Biopredic International, Rennes, 프랑스)을 검정 완충액(NaCl, 트리스-HCl pH = 7.4)에 용해된 이소형 대조군 IgG 또는 항-PAI-1 항체와 함께 항온배양하였다. 상온에서 15분 간 항온배양한 후, 인간 당화 PAI-1(GLYHPAI-A, Molecular Innovation)을 3 nM의 최종 농도까지 첨가하였고 추가 10분 간 더 항온배양하였다. 그런 다음, t-PA(sctPA, Molecular Innovation)를 1 nM의 최종 농도까지 첨가하였다. 응괴 형성을 칼슘 검정 완충액(CaCl2) 중 7.5 mM의 최종 농도까지 희석한 조직 인자(Innovin®, Siemens Healthcare Diagnostics, Marburg, 독일)를 포함하는 활성화 믹스에 의해 유도하였다.
340 nm에서의 흡광도의 역학 판독을 iEMS 마이크로플레이트 판독기(ThermoFischer) 또는 SpectrostarNano(BMG Labtech)를 사용하여 5시간 동안 매 30초마다 수행하였다. 응괴 용해에 대한 효과를 정량하기 위해, 응괴 형성과 응괴 용해 사이의 균형을 반영하는 곡선 하 면적(AUC)을 GraphPad Prism 소프트웨어를 사용하여 계산하였다. 항체 처리 후 응괴 용해의 회복을 아래의 계산식에 따라 결정하였다:
IC50 및 Imax를 Biostat 스피드 소프트웨어를 사용하여 계산하였다.
1 nM 농도의 t-PA는 2시간 내에 정상 혈장의 완전한 용해를 초래하였다. 3 nM-농도의 PAI-1는 t-PA-유도된 응괴 용해를 저해하였다. t-PA 또는 PAI-1 단독의 추가는 응괴 형성에 영향을 미치지 않았다. t-PA 또는 PAI-1 중 어떤 것의 추가도 응괴 형성에 영향을 미치지 않았다.
A44V11 항-PAI-1 항체는 인간 혈소판 부족 혈장 응괴 용해(도 21 참조)를 회복시켰지만, 이소형 IgG1는 그렇지 않았다(도 22 참조). A44V11는 100 nM에서 103%의 Imax와 2 nM의 IC50을 나타냈다(도 23 참조).
APG 항-PAI-1 항체의 인간화 변이체도, 또한, 인간 혈소판 부족 혈장 응괴 용해를 회복하였다(도 24 참조). APGv2는 100 nM에서 114%의 Imax 및 2.1 nM의 IC50을 나타내었다. APGv4는 100 nM에서 116%의 Imax 및 2.8 nM의 IC50을 나타내었다(도 25 참조). 응괴 용해 데이터는 아래의 표 33에 요약되어 있다.
실시예
17: 일차 인간 폐 세포 내
PAI
-1의 A44V11 중화의 평가
PAI-1의 중화에 대한 항체 A44V11의 효과를 폐 세포 기반 시스템에서 조사하였다. TGFβ는 가장 강하고 아주 흔한 전섬유형성 사이토카인으로 간주된다. TGFβ는 배양된 뮤라인 배아 섬유아세포(NIH3T3 세포)에서 PAI-1 발현을 유도하고 t-PA 및 플라스민의 활성뿐 아니라 콜라겐 분해를 저해하는 것으로 보여진다. 문헌(Liu, R-M. Antioxid Redox Signal. 10(2): 303-319(2008))을 참조한다. ATCC(Manassas, Virginia)로부터의 일차 폐 섬유아세포주 LL29(CCL-134) 및 LL97A(CCL-191)를 12-웰 플레이트에 웰 당 200,000 세포의 농도로 밤새 평판배양하였다. 세포를 A44V11 항체 또는 이소형 대조군(IgG)과 5 ng/ml 농도의 TGFβ(R&D Systems, Minneapolis, Minn., cat. #100-B-001)와 함께 48시간 동안 항온배양하였다. 48시간 후, 세포 상층액을 수확하였고 토끼 pAb 항 PAI-1(abcam, ab66705)을 이용한 PAI-1 형태의 검출을 위한 웨스턴 블랏으로 분석하였다.
TGFβ 자극 후 A44V11 항체로 처리된 세포는 PAI-1 밴드를 PAI-1의 절단된 형태에 상응하는 이중항으로 나타냈다(도 26, 레인 5 참조). 대조군 IgG로 처리된 세포는 이러한 이중항 형성을 보이지 않았다(도 26, 레인 6 참조). 본 연구는 A44V11로의 일차 인간 폐 세포의 처리가 PAI-1이 프로테아제에 의해 절단되는 것을 허용하는 내인성 PAI-1 기질 형태를 유도한다는 것을 입증하였다.
실시예
18:
A44V11은
MMP의
활성을
증가시킨다
플라스민은 콜라겐을 포함하는 대부분의 ECM 단백질(섬유증 조직의 주요 단백질성 구성요소)을 분해시킬 수 있는 효소인 MMP을 활성화시킬 수 있다. 이러한 관점에서, 플라스민은 종종 MMP의 일반적인 활성인자로 언급된다. (Loskutoff, et al. J. Clin . Invest. 106(12):1441-43(2000) 참조). PAI-1은 섬유아세포 아폽토시스의 저해가 뒤따르는 플라스민 생성을 차단함으로써 MMP 활성화 및 기질 분해를 감소시킨다. MMP의 활성화를 자극할 수 있는 A44v11의 능력을 폐 세포 기반 시스템에서 조사하였다. ATCC(Manassas, Virginia)로부터의 일차 폐 섬유아세포 LL29(CCL-134) 및 LL97A(CCL-191)를 웰 당 250,000 세포의 농도로 12-웰 플레이트에서 밤새 평판배양하였다. 세포를 A44V11 또는 이소형 대조군(IgG) 및 0.1 μM의 농도의 Lys-플라스미노겐(Molecular Innovation, cat. # HGPG-712)과 48시간 동안 항온배양시켰다. 48시간 후, 세포 상층액을 수확하였고 다양한 종류의 MMP(예를 들어, MMP-1, 2, 3, 7, 8, 9, 12, 13, 및 14를 포함함)의 활성을 제조자의 지시에 따라 Sensolyte 520 generic MMP 검정 키트(AnaSpec, Fremont, CA, cat. # 71158)를 사용하여 검출하였다.
도 27에 나타낸 바와 같이, A44V11은 인간 폐 섬유아세포 내 플라스민-의존성 MMP의 활성화를 자극한다. 이러한 차트는 2개의 대표적인 별도의 실험을 보여준다. A44V11 및 플라스미노겐으로 처리된 세포는 음성 IgG1 항체로 처리된 세포와 비교할 때 실질적으로 증가된 활성화를 나타내었다. 이러한 연구는 A44V11가 플라스민 매개 현상에서 MMP 활성화를 자극한다는 것을 입증하였다.
실시예
19 폐 섬유아세포 마우스 모델(
블레오마이신
챌린지
)에서
A44V11
효능의 분석
블레오마이신에 의해 유도된 실험적인 폐 섬유증은 광범위한 문헌에 의해 지지되는 섬유조직성장의 잘 연구된 모델이다. 폐 섬유증의 이러한 모델은 인간에서 나타나는 섬유증과 닮았고, 기본적인 연구뿐 아니라 잠재적인 치료제의 효과를 산정하는데에도 사용되고 있다. (예를 들어, Molina-Molina et al. Thorax 61:604-610(2006)참조).
블레오마이신
처리된
마우스(섬유증 모델)내
약동력학
연구
인간 PAI-1를 발현하는 유전자이식 마우스(인간화 PAI-1 유전자이식 마우스)를, 마우스 PAI-1(SERPINE1) 유전자 CDS(엑손 및 인트론)(NCBI Ref. No. NM_008871)를 C57BL/6 x 129 마우스(The Jackson Laboratory, Bar Harbor, Maine) 내 대조군의 내인성 마우스 PAI-1 유전자 조절 서열의 조절 하에 상응하는 인간 야생형 PAI-1 유전자 CDS(NCBI Ref. No. NM_000602.3; NC_000007.13)로 교체하여 생성하였다(Klinger, K.W. et al. Proc . Natl . Acad . Sci. USA 84:8548(1987) 참조). 분자 클로닝 및 유전자이식 마우스의 생성을 통상적인 기술 및 제조자 및 사육자 지시에 따라 수행한다. 인간 PAI-1의 발현 및 마우스 PAI-1의 미발현을 동종접합 마우스 내에서 확인하였다. mRNA 및 단백질 수준을, 각각, 표준 qPCR 및 ELISA에 의해 확인하였다. 암컷 동종접합 인간화 PAI-1 유전자이식 마우스(8 내지 9주령 및 22~25 g 중량)을 이러한 절차를 위해 사용하였다. 설치류 사료 및 물은 임의로 제공되었다.
마우스는 0.9% NaCl에 용해된 50 μl의 블레오마이신®(Sanofi, 프랑스)을 2 mg/kg의 용량으로 미세분무기를 통한 기관 내 주사에 의해 받았다. 대조군 마우스는 50 μl의 0.9% NaCl을 받았다. 이러한 절차를 통해, 마우스를 흡입에 의해 이소플루란(isoflurane, TEM, Lormont, 프랑스)으로 마취시키고, 그런 다음 18G 카눌라를 삽입하였다. 마취를 유지하기 위해 카놀라를 산소/이소플루란 혼합물이 공급되는 환풍기에 연결하였다. 마취 후, 미세분무기를 폐에 직접적으로 블레오마이신을 주사하기 위한 카눌라에 도입하였다. 그런 다음, 마우스에서 관상 기관을 없앴고, 마취로부터 회복되도록 하였다. 4일 째에, 3개의 군으로 무작위화한 후, 마우스를 일단 PBS(1 mg/ml) 중 10 mg/kg의 음성 대조군 마우스 IgG1 또는 A44v11의 복강 내 투여로 처리하였다.
블레오마이신 챌린지 후, 지정된 시점(7일 또는 9일)에, 마우스를 자일렌/케타민 믹스로 마취시켰고 개흉하여 안락사시켰다. 시트레이트로 코팅된 튜브 상에서 심장내 수확에 의해 혈액 수집을 수행하였다. 좌측 기관지를 고정하였고, 좌측 폐를 제거하고, 조직학적 분석을 위해 제어되는 압력 하에 고정액(FineFix®, Leica Biosystems, Buffalo Grove, IL)으로 고정하였다. 그런 다음, 카눌라를 기관지폐포요법(BAL) 절차(0.5 ml의 3회 주사에서 주사되고 수확된 1.5 ml의 0.9% NaCl)를 위해 기관에 넣었다. 그런 다음, 단백질 분석을 위해 우측 폐의 4개의 엽을 수확하고, 2조각으로 절단하고, 용해하였다. 모든 실험은 유럽 윤리 로(European ethical lows)에 따라 수행되었고, 국제 윤리 위원회(internal ethical comity, CEPAL, sanofi)에 의해 승인받았다.
A44V11 수준은 코팅된 비오틴화 인간 PAI-1 플레이트를 이용한 ELISA(Molecular Innovation, cat. # HPAIKT)로 결정되었고, 이차 항 마우스 IgG 술포-태그로 표지(MesoScale Discovery, Gaithersburg, Maryland)를 사용하여 검출하였다. 7일에 A44V11으로 처리된 마우스의 경우, 결과는 혈장 중 200 nM, BALF 중 11 nM 및 폐 용해물 중 12 nM였다.
도 28에 나타낸 바와 같이, 4일째에 단일 복강내 용량(10 mg/kg)의 A44V11의 투여는 블레오마이신 챌린지 7일 후 희생된 동물내, BAL 유체 및 폐 용해물에서 거의 완전한 인간 활성 PAI-1의 저해를 달성하였다. 9일 동물의 경우, A44V11(10 mg/kg)은 폐 용해물에서는 거의 완전한 인간 활성 PAI-1의 저해를 달성하였지만, BALF에서는 단지 부분적인 저해만 달성하였다.
섬유소 분해 산물인 D-이량체를 측정하여 섬유소 분해의 정도를 평가할 수 있다. 섬유소 분해를 측정하기 위해, BALF 중 D-이량체의 수준을 제조자의 지시에 따라 ELISA(Asserachrom D-Di, Diagnostica Stago, Asnieres, 프랑스)로 검출하였다. IgG1 음성 대조군과 비교하여, A44V11로 처리된 군의 BALF 중 D-이량체 수준은 7일에서는 대략 2.8배 그리고 9일에서는 대략 1.6배 증가하였는데, A44V11 처리가 섬유소 분해를 증가시킨다는 것을 시사한다(도 29 참조).
블레오마이신으로 챌린지된 마우스 폐에서 섬유증을 감소시키는 A44V11 활성을 더 평가하기 위한 추가적인 연구가 수행되었다. 이러한 연구에서, 마우스는, 연구 기간 길이가 블레오마이신 챌린지로부터 21일이고, 항체(A44V11 또는 IgG1 대조군 항체(10 mg/kg))로의 처리가 4일부터 시작하여 20일까지 3일마다 반복되었다는 것을 제외하고 전술된 약동력학적 연구와 유사한 프로토콜의 대상이 되었다. 블레오마이신 챌린지 21일 후, 동물을 전술된 바와 같이 희생시켰다.
폐 중량에서의 증가는 증가된 섬유증의 지표인 것으로 알려져 있다. 섬유증의 척도로서, 우측 폐 중량을 모든 실험군 내 마우스에 대하여 결정하였다. 도 30에 나타낸 바와 같이, 블레오마이신 점적 주입은 우측 폐 중량의 증가를 유도하였고, 10 mg/kg의 A44V11 항체의 반복되는 투여로 부분적으로 저해되었다. IgG1 음성 대조군 항체를 사용한 반복적인 투여는 블레오마이신 챌린지에 기인한 우측 폐 중량의 증가를 저해하지 않았다. A44V11로 처리된 마우스에서 블레오마이신으로 유도된 우측 폐 중량 증가에서의 감소는 IgG1 음성 대조군 항체로 처리된 유사한 블레오마이신으로 유도된 마우스와 비교할 때 통계적으로 유의하였다(p<0.001). 통계적 분석을 일원분산분석으로 수행하였고 뉴만-켈루스 검정(Newman-Keuls test)이 이어졌다. 이러한 결과는 A44V11이 인간화 PAI-1 마우스 폐에서 블레오마이신으로 유도된 섬유증을 저해하는 반면, 대조군 IgG1 항체는 그렇지 않다는 것을 나타냈다.
폐 내 콜라겐 축적은 섬유증의 또 다른 알려진 지표이다. 콜라겐 축적을 검정하기 위해, 21일에 희생된 마우스로부터 폐 조직을 준비하고, HPLC로 분리하고, 하이드록시프롤린을 측정하였다. 이러한 기술은 다른 곳, 예를 들어, 문헌(Hattori, et al. J Clin Invest. 106(11):1341-1350(2000))에 상세히 기술되어 있다. 간략하게, 폐 조직을 105℃에서 22시간 동안 산성 조건(6M HCl) 하에 가수분해하여 제조한 후, 증발시켰다. 일차 아민을 OPA(프탈알데하이드)에 의해 폐 조직에서 차단하였고, 프롤린/하이드록시프롤린을 NBD(4-클로로-7-니트로벤조퓨라잔)(Santa Cruz Biotech., Santa Cruz, CA)를 사용하여 특이적으로 표지하였다. 그런 다음, 가수분해물을 아세토니트릴 구배 하에 HPLC(Shimazu Corp., Kyoto, 일본)를 사용하여, Synergi™ 4 μm 하이드로-RP 80 Å, LC 컬럼 150 x 3 mm 컬럼(Phenomenex, Torrance, CA, cat. # 00F-4375-Y0)에서 분리하였다. 알려진 양의 하이드록시프롤린의 표준 곡선이 피크(들)를 정량하기 위한 참조로 사용하였다. 대표적인 정량 데이터가 도 31에 나타나 있다.
하이드록시프롤린 함량에 의해 검출된 폐 콜라겐 축적은 블레오마이신으로 챌린지된 동물에서 증가하였다. 폐 콜라겐 축적에서의 이러한 증가는 10 mg/kg으로의 A44V11 항체의 반복되는 투여에 의해 통계적으로 감소하였다(p<0.08). (도 31 참조). IgG1 음성 대조군 항체를 이용한 반복되는 투여는 블레오마이신 챌린지에 기인한 폐 콜라겐 축적의 증가를 저해하지 않았다. A44V11로 처리된 마우스에서, 블레오마이신으로 유도된 콜라겐 축적 증가의 감소는 IgG1 음성 대조군 항체로 처리된 유사한 블레오마이신으로 유도된 마우스와 비교하여 통계적으로 유의하였다(p<0.05). A44V11로 처리된 마우스는 IgG1 대조군으로 처리된 마우스보다 대략 44% 미만의 콜라겐 축적의 증가를 보였다.
실시예
20: 원숭이
LPS
챌린지
모델에서
A44V11
활성의 평가
원숭이에서 급성 지질다당류(lipopolysaccharide, LPS) 챌린지 모델을 생체 내 A44V11의 PAI-1 중화 효능을 결정하기 위해 사용하였다. LPS 챌린지 모델은 문헌(Hattori, et al. J Clin Invest. 106(11):1341-1350(2000))에 기술되어 있다. 원숭이 혈장 및 간 시료 내 PAI-1에 대한 A44V11 mAb 의 활성을 평가하였다. 구체적으로, 실험은 A44V11(5 mg/kg, IP) 또는 IgG1(음성 대조군, 5 mg/kg, 복강 내 투여)로 사전 처리(24시간 전)된 마취된 원숭이에서 PAI-1의 혈장 및 조직 수준에 대한 고용량의 LPS(100 μg/kg - IV)의 영향을 평가하도록 설계되었다. 실험은 유럽 윤리 로(European ethical lows)에 따라 수행하였고 국제 윤리 위원회(CEPAL, sanofi)에 의해 승인받았다.
4 내지 9 kg의 중량의 시노몰구스 필리핀 원숭이(Macaca fascicularis)(수컷 및 암컷)를, 0.12 내지 0.16 mL/kg으로의 Zoletil 50(Virbac, Taguig City, 필리핀)으로의 IM 유도 후 공기/산소 및 이소플루란(1 내지 3%)의 기체 믹스의 흡입을 포함하는, 장기간 마취(적어도 8시간) 전 밤새 금식시켰다. 원숭이 체온을 가열 패드를 이용하여 생리학적 범위 내로 유지시켰다. 카테터 삽입 후, LPS(혈청형 0127~B8)를 100 μg/kg(0.4 mL/kg)의 용량으로 부요측피정맥 내에 1분 볼루스로 투여하였다. 다양한 시점에서, 혈액 시료 및 간 시료를 채취하였다. 혈액 시료(시트레이트/EDTA 상)를 수확하고 원심분리하여 혈소판 부족 혈장을 단리하였다. 간 생검물 및 말단 부검을 -80℃에 저장하였다.
활성 PAI-1, D-이량체 및 플라스민-α2 항플라스민 수준을 제조자의 지시에 따라 상업적으로 이용가능한 ELISA 검정(Mol. Innovation, cat. # HPAIKT; Asserachrom D-이량체; 플라스민-A2 항플라스민, Diagnostica Stago)을 사용하여 결정하였다.
혈장에서, 활성 PAI-1 수준은 A44v11이 투여된 모든 원숭이에서 약 30 ng/ml에서 10 ng/ml 미만으로 감소하였다. (도 32(a) 참조). LPS 투여(100 ug/kg) 후 활성 PAI-1 수준에서의 증가는 없었다. (도 32(a) 참조). 반대로, 음성 IgG1 대조군으로 처리된 원숭이는 LPS 투여 후 활성 PAI-1 수준에서 강한 증가를 보였는데, 약 4시간(대략 50 내지 약 250 ng/ml)에서 최대로 발생하였다. (도 32(b)참조). 따라서, 음성 IgG1 대조군으로의 처리는 LPS 투여 후 강하게 증가한 혈장 내 활성 PAI-1 수준을 감소시키지 않는다. (도 32(b) 참조).
간 생검물 용해에서, 유사한 현상이 관찰되었다. A44V11 mAb로 처리된 원숭이는 LPS 처리 후 활성 PAI-1 수준에서 증가를 보이지 않았다. (도 33(a) 참조). 반대로, LPS 투여는 음성 IgG1 대조군으로 처리된 원숭이로부터의 간 생검물 용해물에서 활성 PAI-1(최대 3 ng/mg)의 강한 증가를 유도하였다(도 33(b) 참조).
PAI-1 중화와 동시에, A44V11으로 처리된 원숭이 내 D-이량체 수준(도 34(a)참조)은 음성 IgG 대조군으로 처리된 원숭이(도 34(b) 참조)보다 일반적으로 높은 것으로 나타났으며, 따라서 원숭이에서 A44V11 처리도 또한 혈장 내 섬유소 분해의 증가를 유도한다는 것을 시사한다.
마지막으로, A44V11으로 처리된 원숭이의 혈장 시료는 음성 IgG 대조군으로 처리된 원숭이에서의 PAP 수준에 비하여 증가된 수준의 플라스민-α2 항플라스민(PAP) 복합체를 나타내었다(도 35(a) 및 (b) 참조). A44V11의 존재 하에 PAP 복합체 및 D-이량체의 이러한 증가는 플라스민 생성에서의 증가를 나타낸다.
실시예
21: 복부 유착 마우스 모델에서의
A44V11
활성의 평가
수술적 손상의 마우스 자궁각 모델에서 유착의 형성에 대한 항-PAI-1 항체 A44V11로의 처리의 효과를 평가하였다. 마우스 자궁각 접합 및 전기소작기 절차는 장막 표면을 파괴하여, 자궁 조직에 열 손상을 유발하고, 치유 과정 중 손상된 조직 표면을 접합하는데, 이는 100%의 미처리 동물에서 궁극적으로 수술 후 유착을 초래한다. 모델 및 수술 절차는 이전에 문헌(Haney A.F. et al.,(1993). Fertility and Sterility, 60(3): 550-558)에 기술되어 있다.
이러한 유착 연구를 위해, 인간화 PAI-1 이식유전자를 발현하는 위에서 생성된 유전자이식 암컷 마우스(대략 9주령, 대략 20 g 중량)를 사용하였다. 42 마리의 성숙 유전자이식 암컷 마우스를 2개의 군으로 나누었고, 문헌(Haney A.F. et al.,(1993))에 상세히 기술된 바와 같이 자궁각(UH) 사이에 유착을 생성하도록 설계된 수술 절차를 거쳤다. 간략하게, 수술을 위해 각각의 동물을 IACUC 가이드라인에 따라 이소플루란으로 마취시켰고, 일상적인 중심선 개복(대략 1.0 cm 미골에서부터 겸상 돌기)을 수행하였다. UH를 확인하였고, 각각의 뿔의 근육벽을 통해 조심스럽게 넣은 단일 7-0 프로렌(Prolene) 봉합사(Ethicon Inc., Somerville, N.J)로 안쪽으로 접합시켰고, 뿔들을 자궁관 접합에서의 난관의 접합점 바로 아래에서 묶었다. 난소 혈관 공급을 손상시키지 않도록 주의하였다. 전기소작기 손상을 유도하기 위해, 대략 2x6 mm의 면적을 덮은, 각각의 자궁각의 내측 표면에 양극성 전기소작기 단위를 사용하였다(Valley Lab Surgistat, 고체상 전기수술 단위, 모델 No. B-20). 전기소작기 단위는 다음과 같이 설정되었다: 볼트 100, 130 Hz, 50~60 Amps. 3 mm 넓이의 소작기 팁을 3의 설정의 순수한 응고 전류로 사용하였고, 동력 공급을 개시하였고, 뿔 당 2개의 화상 스팟을 위해 1초 간 조직을 터치하였다. 근육 절개를 연속 봉합 방식으로 5-0 Vicryl, BV-1 테이퍼 니들(Ethicon Inc.)을 이용하여 덮었다. 피부를 수평 매트리스 봉합 패턴으로 5-0 프로렌, BV-1 테이퍼 니들(Ethicon Inc.)로 덮었다.
UH 손상의 생성 후, 군 1 동물을, 소작기 화상에 적용되는, 0.16 ml의 부피의 이소형 대조군 항체(30 mg/kg)로 처리하였다. 군 2 동물을 동일한 방식으로 0.16 ml의 부피의 A44V11 항체(30 mg/kg)로 처리하였다. 각 군에서, 동물을 6시간(n=5), 72시간(n=4), 또는 7일(n=12)에 안락사시켰다. (아래 표 34 참조). 72시간 및 7일에 안락사가 계획된 동물은 수술 48시간 후 복강 내 주사(IP)로 주사된 제2 용량의 항체(30mg/kg)를 받았다.
주의: 모든 동물은 치료에 앞서 형성된 소각기 화상 및 봉합에 의해 접합된 자궁각을 가졌다.
효능 평가 및 분석:
동물을 지시된 시점에 안락사시켰고, 유착의 형성을 평가하였다. 간략하게, 자궁 분기에서부터 난관 바로 아래 위치한 접합 봉합부까지의 뿔의 길이를 측정하였다. 자궁각을 둘러싸는 2개의 외부 봉합체를 제거하였고 자궁각 사이의 유착의 길이를 현미경의 도움으로 측정하고, 문서화하고, 존재 또는 부재(예/아니오)로 기록하였다. 또한, 유착 형성에 관련된 임의의 조직이 기록될 것이지만, 유착 면적의 길이에는 포함되지 않을 수 있다. 자궁각 사이의 유착 길이의 평균 백분율의 분포를 Shapiro-Wilk 검정을 사용하여 정규성에 대하여 점검하였다. 정규 분포한다면 Tukey Kramer 분석을 사용하고, 정규 분포하지 않는다면 Wilcoxon Rank-Sum 분석을 사용하여 군들을 서로 비교하였다. 모든 경우에서, p-값 ≤ 0.05은 통계적으로 유의한 것으로 간주되었다. A44V11으로 처리된 동물은 접합된 자궁각 사이에서 유의하게 낮은 백분율의 유착 형성 길이를 나타내었다(표 35 참조).
*p= Wilcoxon Rank Sum 분석, 카이 제곱 추정에 의해 이소형 대조군에 대해 통계적으로 유의함
활성
PAI
-1 및 tPA 수준의 검출
안락사 후, 동물은 평가를 위해 수집된 혈액(혈장), 복강 내 유체(IPF), 및 자궁각 시료를 가졌다. 시료의 수집은 통상적인 기술을 사용하여 수행되었다. 혈장, IPF, 및 자궁각 시료에서 활성 PAI-1 및 tPA 수준을 ELISA(인간 PAI-1 활성 ELISA 키트, Cat. # HPAIKT, Molecular Innovations, Novi, MI)를 사용하여 평가하였다. 데이터를 엑셀, JMP, 및 프리즘 그래프 패드 소프트웨어를 사용하여 가공하였다. 모든 경우, p-값 ≤ 0.05을 통계적으로 유의한 것으로 간주하였다. 6시간 및 7일 시점에서, 감소된 수준의 활성 PAI-1이 A44V11 대 이소형 대조군으로 처리된 동물에서 IP 유체 및 자궁각 용해물 내에서 발견되었다. (도 36 참조). 6시간에서의 IPF 내 감소된 수준의 활성 PAI-1은 이소형 대조군에 대한 A44로 처리된 동물에서 6시간 시점에서 IP 유체에서 보여지는 통계적으로 유의한 결과였다(독립표본 t-검정에 의해 p<0.001).
실시예
22: 인간화된 항체
A44V11의
결정 구조
Fab
A44V11의
발현 및 정제
재조합 Fab(rFab)를 경쇄 또는 C-말단 His-태그 중쇄를 암호화하는 2종의 플라스미드를 이용하여 일시적으로 형질감염된 HEK293 세포로부터 수득하였다. 원심분리 및 여과 후, 세포 상층액으로부터의 rFab를 고정화 금속 친화도 수지에 적용하였다. 수지로부터 용리된 후, rFab를 PBS에 대하여 집중적으로 투석하고 4℃에 저장하였다.
시노몰구스
또는 시노
PAI
-
1으로
지칭되는 필리핀 원숭이
PAI
-1의 공급원:
재조합 성숙 시노몰구스 PAI-1(24-402)을 E. coli 내에서 봉입체로 발현시켰고 재조합 단백질을 통상적인 방법을 사용하여 정제하였다.
인간
PAI
-1의 공급원:
재조합 성숙 인간 PAI-1(24-402)을 Molecular Innovations Inc.(카탈로그 번호 CPAI)로부터 구입하였다. 재조합 성숙 인간 PAI-1을 Berkenpas 등(1995, EMBO J., 14, 2969-2977)에 의해 기술된 돌연변이(N150H, K154T, Q319L, M354I)를 삽입함으로써 활성 형태로 안정화시켰다.
복합체의 제조 및 정제:
재조합 Fab 및 항원을 1.5:1 몰 비로 혼합하고, 상온에서 30분 간 항온배양하였고, 복합체를 25 mM MES pH 6.5, 150 mM NaCl로 평형화시킨 Superdex 200 PG 컬럼(GE Healthcare) 상 분취 크기 배제로 더 정제하였다.
Fab
A44V11
+ 시노
PAI
-1 복합체의 결정화
복합체를 25 mM MES pH 6.5, 150 mM NaCl 중 10 mg/ml로 농축하였다. 복합체를 16~24% 에탄올, 100 mM Tris pH 8.5 중에 결정화하였다. 에틸렌 글리콜(30%)을 동결보호제로 사용하였다. 결정은 ESRF의 ID29 빔라인 상 공간 그룹 P321(a=b=193 Å, c=144 Å) 내에서 약 3.3 Å으로 회절되었다. 데이터를 XDS 및 Scala(GlobalPhasing Ltd., Cambridge, UK)의 조합으로 가공하였다
복합체
Fab
A44V11
/시노-
PAI
-1의 구조 결정:
Fab 가변 도메인의 모델을 Maestro에서 Prime(Schrodinger, New York, NY)을 사용하여 제작하였다. 불변 도메인을 공개된 구조 3FO2로부터 얻었다. 2종의 상이한 모델의 인간 PAI-1을 사용하였다: 잠복형을 1LJ5로부터, 활성 형태를 1OC0으로부터 얻었다. 매튜 상관계수(Matthews Coefficient, VM , 단백질 분자량의 단위 당 결정 부피)의 계산은 비대칭 단위 내 최대 4개의 복합체가 존재한다는 것을 시사한다(VM 2. 2는 90 킬로달톤(KD)의 복합체 크기를 가정함). Phaser(CCP4 suite)(McCoy, et al. J. Appl . Cryst. 40: 658-674(2007)를 사용하여 분자 교체를 수행하였고, 2종의 단량체의 잠복성 PAI-1 및 2종의 가변성 Fab 도메인을 확인하였다. 불변 도메인에 대한 추가적인 밀도가 명확하게 시각화되었는데, 이는 수동으로 위치시켜야만 한다. 4.3(71% 용매)의 VM에 상응하는 용액이 또한 패킹 일관성을 조심스럽게 평가하였다. 구조를 비결정학 대칭성을 사용하는 Buster(GlobalPhasing)로 29.2%(R인자 25.8%)의 Rfree로 개량하였다. 불변 도메인은 결정화 패킹으로 안정화되지 않으며, 전자 밀도 맵에서 불량하게 해결된다.
Fab
A44V11
+ 인간
PAI
-1 복합체의 결정화
단백질 결정화는 x-선 결정학 방법에 의한 생물분자 구조 결정의 병목이다. 단백질 결정화의 성공은 결정화 실험에서 사용되는 단백질 분자의 품질에 직접적으로 비례하는데 가장 중요한 품질 기준은 용액 내 단백질의 순도 및 (분자적 및 형태적) 균질성이다.
우선, PAI-1/Fab mAb 복합체 구조를 결정하기 위해, 원래의 mAb A44를 사용하여 파파인 분해로 Fab 단편을 제조하였다. 이러한 Fab의 대규모 제조는 인간 야생형(wt) PAI-1 단백질과의 복합체 내에서 복합체화되고 정제된 이종 Fab 단편을 초래하였다. 얻어진 단백질 복합체를 7 mg/ml 농도까지 농축하였고, 2가지의 상이한 온도(4℃ 및 19℃)에서 800개의 개별적인 결정화 조건에서 결정화하기 위해 스크리닝하였다. 어떠한 결정화 히트도 검출되지 않았다. 단백질 복합체 균질성을 개선하기 위해, 재조합 6-His 태그 Fab A44를 생산하고, 정제하고, 인간 야생형 PAI-1 단백질과 복합체화하였다(도 36 참조).
복합체 결정화 스크리닝으로 20%PEG10K + 0.1M 아세트산 나트륨 pH4.6 조건 하에 제1 결정화 히트를 얻었다. 통상적인 결정화 방법인 Microseed Matrix Seeding에 의한 결정화 최적화 및 원위치(in situ) 트립신 용해 결정화는 결정의 품질을 유의하게 개선하지 않았다. 최적 수득 결정은 침상이었고, 구조 결정에 충분치 않는 해상도(10 Å)로 x-선을 회절시켰다.
복합체 결정의 결정화의 실패는 복합체 구조적 이종성에 의해 잠재적으로 설명될 수 있다. 야생형 PAI-1 분자는 3종의 구분되는 형태(활성, 잠복성, 및 기질)를 채용하는 것으로 알려져 있는데, 이는 결정화를 방해할 수 있다. 결정의 품질을 개선하기 위해, 잠복성 PAI-1 복합체화된 6-His 태그 A44 Fab를 생성하였다. (도 37 참조).
상응하는 복합체를 생성하였고, 6-His 태그 Fab A44/wt PAI-1 단백질 복합체에 앞서 사용된 조건 하에 드 노보 결정화에 대해 스크리닝하였다. 시험된 1000가지를 초과하는 조건 중 오직 결정화 히트만이 20% PEG3350+0.2M nH4 아세테이트 +4% MPD + 50 mM Mes pH6 조건하에 복합체를 확인하였다(도 39(a) 참조). 집중적인 최적화 후, 3D 결정이 얻어졌다. 싱크로트론 고강도 X-선 빔을 사용한 회절 시험은 어떠한 회절 징후도 나타내지 않았다(도 39(b) 참조, 대표적인 최적화된 결정을 도시함).
단백질의 일부분의 유연성을 낮추기 위해 A44 Fab 단편을 재조합적이지만 앞서 사용된 6-His 태그와 같은 인공적인 태그를 사용하여 않고 생산하기로 결정하였다. 성공적인 결정화의 기회를 더 높이기 위해, PAI-1(N150H, K154T, Q319L, M354I)의 활성 형태 돌연변이를 Molecular Innovations(Cat. #CPAI, Novi, MI)으로부터 구입하였고 인공적인 태그가 결여된 Fab A44 단백질를 가지는 복합체의 제조에 사용하였다. 복합체를 25 mM MES pH 6.5, 150 mM NaCl에 12 mg/ml까지 농축하였다. 허용가능한 막대형 단일 결정이 10% PEG3350, 100 mM 황산 암모늄에서 수득되었고, 30% 에틸 글리콜(도 40 참조)의 첨가에 의해 동결보존되었다. 이러한 결정을 3.7 Å 해상도까지 회절시켰고, 집중적인 동결건조 최적화 후, 구조 결정에 적합한 x-선 회절 데이터 세트가 얻어졌다(3.3 Å). 데이터세트를 신크로트론 SOLEIL(Saint-Aubin, 프랑스)의 빔라인 Proxima 1에서 3.3 Å까지 수집하였다. 공간그룹은 P212121(a=105, b=152 c=298)이다. 데이터를 XDSme 스크립트(XDS ref, Xdsme ref)를 사용하여 가공하였다.
복합체
Fab
A44V11
/인간-
PAI
-1의 구조 결정:
뚜렷하지 않은 것(CCP4)은 공간그룹 식별에서 단지 40% 신뢰도만을 나타낸다. 따라서, P222 포인트 그룹의 모든 가능한 공간 그룹 변이체를 시험하기 위해 초기 분자 교체를 Amore(CCP4)로 수행하였다: P212121가 분명하게 확인되었다. Phaser(Phaser, CCP4)를 이용한 최종 분자 교체는 비대칭 단위 내 Fab의 활성 PAI-1/가변 도메인 4개의 이량체를 확인하였다. 불변 도메인을 전자 밀도 맵에 수동으로 첨가하였다. 구조를 비결정학 대칭을 사용한 Buster(GlobalPhasing)로, 28%(R인자 24.1%)의 Rfree까지 개량하였다.
에피토프
및
파라토프
구조 분석
에피토프 및 파라토프 영역을 시노 및 인간 복합체에서 형성된 바와 같이 확인하였고, 복합체를 비교하였다. 결정 구조를 인간 및 시노 PAI-1와의 복합체 내 A44V11에 대해 3.3 Å까지 결정하였다. 두 구조의 중첩(도 40)은 A44V11의 파라토프가 PAI-1의 잠복성 및 활성 헝태와 유사하다는 것을 보여준다. Fab A44는 인간 PAI-1의 활성 형태 및 시노 PAI-1의 잠복성 형태를 인식하였다. 도 42는 활성 인간 PAI-1(도 42(a)), 및 잠복성 시노 PAI-1(도 42(b))에서 Fab A44에 의해 인식되는 PAI-1 에피토프를 도시한 것이다. 파라토프를 인식하는 잠복성 형태는 활성 형태를 인식하는 파라토프의 일부분이다.
PAI-1은 상호작용의 표면적의 분석에서 볼 수 있는 바와 같은 A44V11의 중쇄와 주로 상호작용한다. 활성 인간 PAI-1과 중쇄(4개의 복합체의 평균) 사이의 상호작용의 표면적은 674 Å2이다. 활성 인간 PAI-1과 경쇄(4개의 복합체의 평균) 사이의 상호작용의 표면적은 372 Å2이다. 잠복성 시노 PAI-1과 중쇄(2개의 복합체의 평균) 사이의 상호작용의 표면적은 703 Å2이다. 잠복성 시노 PAI-1과 경쇄(2개의 복합체의 평균)의 상호작용의 표면적은 360 Å2이다. 중쇄 및 경쇄의 파라도프의 도시에 대해서는, 각각, 도 43 및 44를 참조한다.
파라토프의 A44V11 부위의 잔기는 아래 표 36에 나타나 있다. 이탤릭체인 잔기는 활성 PAI-1와의 상호작용에 관련되어 있지만, 잠복성 형태에는 관련되어 있지 않은 반면, 밑줄 친 잔기는 잠복성 형태와만 상호작용한다. 모든 기타 잔기들은 양 계면에 포함된다.
인간 및 시노 PAI-1 분자의 상이한 형태에도 불구하고, 동일한 잔기들이 Fab A44(아래 인간 PAI-1의 서열 내 굵은 글씨 잔기)(서열 번호 1)와의 상호작용에 관련된다:
VHHPPSYVAHLASDFGVRVFQQVAQASKDRNVVFSPYGVASVLAMLQLTTGGETQQQIQAAMGFKIDDKGMAPALRHLYKELMGPWNKDEISTTDAIFVQRDLKLVQGFMPHFFRLFRSTVKQVDFSEVERARFIINDWVKTHTKGMISHLLGTGAVDQLTRLVLVNALYFNGQWKTPFPDSSTHRRLFHKSDGSTVSVPMMAQTNKFNYTEFTTPDGHYYDILELPYHGDTLSMFIAAPYEKEVPLSALTNILSAQLISHWKGNMTRLPRLLVLPKFSLETEVDLRKPLENLGMTDMFRQFQADFTSLSDQEPLHVALALQKVKIEVNESGTVASSSTAVIVSARMAPEEIIIDRPFLFVVRHNPTGTVLFMGQVMEP
인간 PAI-1에 대한 A44V11 결합 에피토프에 대한 약칭은 아래와 같다:
E-X-X-Q(서열 번호 156);
L-X-R (서열 번호 157);
T-D-X-X-R-Q-F-Q-A-D-F-T-X-X-S-D-Q-E-P-L(서열 번호 158)
요약하면, FabA44를 인식하는 PAI-1의 시노 및 인간 에피토프는 양 형태에서 동일하다. Fab A44는 인간 및 시노 PAI-1을 인식하지만, 마우스 또는 랫트 PAI-1을 인식하지 않는다.
실시예
23:
A44V11
특이성 및 교차 반응성의 결정
A44V11의 특이성 및 반응성을 결정하기 위해, A44V11 에피토프의 서열(위 참조)을 사용하여 ScanProsite(SIB Swiss Institute of Bioinformatics) 데이터베이스를 이용한 모티프 검색을 사용한 다른 단백질에서의 유사한 에피토프를 검색하였다. 추가적인 세부사항에 대해서는 문헌(Artimo, P. et al. Nucleic Acids Res. 40(W1):W597-603(2012))을 참조한다. 검색 내 위치한 모든 에피토프 서열 매치는 PAI-1과 관련이 있었는데, 이는 A44V11 항체가 PAI-1에 특이적이라는 것을 시사한다.
A44V11 에피토프를 또한 Med-SuMo에 따라 인실리코 프로파일링 및 분자 모델링을 사용하여 다른 알려진 x-선 구조(3D 검색)에 비교하였는데, 이는, 예를 들어, 수소 결합, 전하, 소수성 및 방향족 기를 포함하는 단백질 표면 상의 생화학적 기능을 검출하고 비교한다. Med-SuMo 분자 모델링은 문헌(Jambon, et al. Bioinformatics 21(20):3929-30(2005))에 더 기술되어 있다. A44V11 에피토프의 3D 검색은 인간 알파-1-안티트립신내 유사한 모티프(AAT1) 에 위치하였다. 그러나, 추가적인 조사 시, AAT1 모티프는 A44V11 에피토프와 유의한 차이를 가지는 것으로 밝혀졌으며, A44V11는 결합할 가능성이 낮다. 따라서, A44V11 에피토프의 서열 패턴 및 3D 패턴 분석은 다른 인간 단백질들과 최소의 교차반응성을 가져야 한다는 것을 시사한다.
A44V11에 대한 인간 및 시노 PAI-1 에피토프를 마우스 및 랫트 PAI-1로부터 제안된 에피토프에 비교하였다. 서열은 서열 번호1(PAI-1 인간), 서열 번호162(PAI-1 시노), 서열 번호163(PAI-1 마우스), 및 서열 번호 164(PAI-1 랫트)로부터 발췌된다. 랫트 및 마우스 PAI-1은 인간 PAI-1와 각각 75% 및 79% 서열 동일성을 가진다. 상이한 PAI-1 서열들의 정렬은 그들 각각의 에피토프에서 랫트/마우스 및 인간/시노 서열 사이에서 유의한 차이를 보이는데, 이는 A44V11가 랫트 또는 마우스 PAI-1을 인식할 가능성이 낮다는 것을 시사한다(도 45 참조). 예를 들어, 마우스 PAI-1 아미노산 Ser300, Thr302, Gln314은 인간/시노 PAI-1 카운터파트와 상이하다. 이러한 잔기에서의 차이는 제안된 에피토프에서의 변화를 나타내며, 마우스 PAI-1는 A44V11에 인식될 수 없다. 마우스 PAI-1과 복합체 인간 PAI-1/A44V11(도 46)의 구조 비교는 A44V11 항체로부터 인간 및 마우스 활성 모두를 얻는 것이 가능하지 않아야 한다는 것을 추가적으로 나타낸다.
A44V11에 대해 확인된 에피토프를 더 검증하기 위해, 인간 및 시노 A44V11 에피토프를 비브로넥틴의 결합 영역과 비교하였다. 비브로넥틴의 소마토메딘 B 도메인과의 복합체 내에서 인간 PAI1의 구조가 공개되어 있다(1OC0). 이러한 2종의 복합체의 구조를 비교하였다(도 47 참조). 구조적 비교는 A44V11의 결합이 비브로넥틴과 PAI-1 상호작용에 영향을 미치지 않을 것이라는 것을 시사한다.
A44V11 에피토프를 다른 공개된 항-PAI1 항체의 에피토프에 비교하였다. 128~156 영역내 잔기에 결합하는 다른 공개된 항-PAI-1 항체 MA-55F4C2 및 MA-33H1와 A44V11 에피토프의 중첩이 발견되지 않았다(Debrock et al. Thromb Haemost, 79:597-601(1998)참조).
마지막으로, A44V11 항체의 특이성 및 교차 반응성의 결여를 비아코어로 확인하였다. A44V11 에피토프의 예측된 고유 서열 및 3D 구조에 기반한 분자 모델링 연구는 A44V11가 인간 및 시노 PAI-1에 특이적이라는 것을 강하게 나타낸다.
실시예
24: 수소/중수소 교환 질량 분광계(
HDX
-MS)에 의한
에피토프
맵핑
각 항체의 에피토프를 더 특성분석하기 위해 질량 분광계(MS)로 모니터링된 수소/중수소 교환(HDX)을 본원에 개시된 PAI-1-결합 항체에 적용하였다. HDX MS는 다양한 상태의 동일한 단백질을 비교하는 데 특히 유용한 기술이다. 상세한 방법론 및 단백질 치료제에 대한 HDX MS의 적용이 문헌(Wei, et al., Drug Discovery Today, 19(1): 95-102(2014))에 개시되어 있다. 간략하게, 수용성의 경우, 모든 H2O 용매는 구분되는 분광학적 성질을 가지는 수소의 동위원소로 교체된 다음, 하나가 이러한 교환 절차를 따를 수 있다. 가장 최근의 HDX 실험의 경우, 중수소화 또는 "중(heavy)" 수(D2O)가 사용된다. 특히, 골격 질소에 결합된 수소(또한, 골격 아미드 수소로도 지칭됨)이 단백질 형태를 탐지하는데 특히 유용하다. 예를 들어, 문헌(Marcsisin, et al. Anal Bioanal Chem . 397(3): 967 - 972(2010))을 참조한다. 단백질의 노출되고 동적인 영역은 빠르게 교체될 것인 반면, 단백질의 보호되고 단단한 영역은 느리게 교체될 것이다. 모든 관련된 조건(pH, 온도, 이온 강도, 등.)은 일정하게 유지됨으로, 구조(용매 접근성, 수소 결합)에서의 차이만이 이러한 교환에 영향을 미칠 것이다. PAI-1와 항체의 상호작용은 항원의 일정한 부위의 표지를 차단할 것임으로, 결합의 자리(에피토프)에 기반한 상이한 판독이 생성된다.
실험 방법:
시노-PAI-1(10 uM), A44v11에 결합한 시노-PAI-1(각 10 μm) 및 APGv2에 결합한 시노-PAI-1(각 10 μm)의 스톡 용액을 PBS, pH 7.2에 제조하였다. 단백질 용액을 1시간 동안 상온에서 항온배양함으로써 결합 평형에 도달하도록 하였다. 50 pM 미만의 Kd 값에 기반하여, 각각의 항체:항원 복합체는 아래에 기술된 표지 조건 하에 99% 초과로 결합하였다.
중수소 교환, ?칭, 및 시료 주입은 자동화된 로봇 시스템(LEAP Tech., Carrboro, NC)으로 처리하였다. 단백질 용액의 분취액을 표지 완충액(99.9% D2O 중 PBS, pD 7.2)으로 10배 희석하였고 10초, 1분, 5분, 또는 4시간 동안 20℃에서 항온배양되도록 하였다. 중수소 교환 시점의 종료시, 50 μL의 표지 용액을 동일한 부피의 미리 차갑게 한(0℃) 100 mM 인산 나트륨, 4 M 구아니딘 염산, 0.5 M TCEP, pH 2.5에 첨가함으로써 표지 반응을 ?칭하였다. 중수소화되지 않은 대조군을 동일한 방식으로 H2O 중 PBS에 10배 희석함으로써 제조하였다.
각각의 ?칭된 시료(50 μL, 50 pmol의 각 단백질)를 HDX Technology를 구비한 Waters nanoAcquity(Waters Corp., Milford, MA)에 즉시 주입하였다. 단백질을 20℃로 유지되는 2.1 mm x 30 mm Enzymate BEH 펩신 컬럼(Waters Corp.)으로 선 상에서 분해하였다. 모든 발색 요소는 초고성능 액체 크로마토그래피(UPLC) 시스템 내의 쿨링 챔버 안에서 0.0 ± 0.1℃로 유지되었다. 얻어진 펩티드를 가두고 3분 100 μL/분으로 3분 간 탈염한 다음, 1.0 x 100.0 mm ACQUITY UPLC HSS T3 컬럼(Waters Corp.)에서 40 μL/분으로 12분 간 2~40% 아세토니트릴:물 구배를 가지고 분리하였다. 역교환에 대한 중수소 수준을 보정하지 않았고, 상대적으로 보고하였다. 모든 비교 실험은 역교환 보정에 대한 필요성을 무력화하는 동일한 조건 하에 수행하였다. 모든 실험은 3반복으로 수행하였다. 주입 사이의 펩티드 잔재를, 각 시행 후 모든 컬럼에 50 μL의 1.5 M 구아니딘 염산, 0.8% 포름산, 및 4% 아세토니트릴을 주입함으로써 제거하였다.
질량 스펙트럼을 HDMSe 모드로 시행되는 표준 전자분무 공급원을 구비한 Waters Synapt G2-Si 기기(Waters Corp.)로 획득하였다. 기기 설정은 다음과 같았다: 모세관은 3.5 kV였고, 샘플링 콘은 30 V였고, 공급원 오프셋은 30 V였고, 공급원 온도는 80℃였고, 탈용매화 온도는 175℃였고, 콘 기체는 50 L/hr였고, 탈용매화 기체는 600 L/h였고, 네뷸라이저 기체는 6.5 bar였다. 질량 스펙트럼을 50~1700의 m/z 범위에 걸쳐 획득하였다. 잠금집단 프로브를 통한 100 fmol/uL 인간 [Glu1]-피브리노펩티드 B 동시 투입에 의해 각 시행에서 질량 정확도를 유지하였다.
중수소화되지 않은 펩신 펩티드의 MSE 확인을 ProteinLynx Global Server 소프트웨어(Waters Corp.)를 사용하여 수행하였다. 각 펩티드에 대한 중수소 흡수를 DynamX 2.0 소프트웨어(Waters Corp.)를 사용하여 결정하였다. 상대적인 중수소 수준을 중수소화되지 않은 펩티드의 동위원소 분포의 중심을 상응하는 중수소 표지된 펩티드의 중심으로부터 감하여 계산하였다. 중수소 흡수 도표를 소프트웨어에 의해 자동으로 생성하였다.
PAI
-1 상태에 대한 중수소 흡수의
모니터링
선상 펩신 분해 후, 95.3% 서열 커버리지를 초래하는 150개의 중첩된 시노-PAI-1 펩신 펩티드를 확인하였다(도 48 참조). 다음의 3가지 상이한 단백질 상태에 대한 중수소 흡수를 150개 펩티드 모두에서 모니터링(10초부터 4시간까지)하였다: (1) 시노-PAI-1 단독; (2) 시노-PAI-1에 결합된 A44v11; 및 (3) 시노-PAI-1에 결합된 APGv2.
대부분의 시노-PAI-1 펩티드는 3가지 상태에서 거의 동일한 중수소 흡수를보였는데, 이는 이러한 영역 내 시노-PAI-1과 mAb 사이에서 상호작용이 없다는 것을 나타낸다. 이러한 결과를 가지는 하나의 대표적인 펩티드 영역(잔기 139~152)을 도시한 도 49(a)를 참조한다. 반대로 잔기 44~64에 삽입된 펩티드는 A44v11 또는 APGv2(도 49(B))에 결합할 때 교환으로부터의 유의한 보호(감소된 중수소 흡수)를 나타냈다. 또한, 잔기 295~322에 삽입된 펩티드도, 또한, A44v11 또는 APGv2(도 49(c))에 결합할 때 교환으로부터의 유의한 보호를 보였다. 이러한 영역에서, 보호의 범위는 시노-PAI-1이 APGv2보다는 A44v11에 결합할 때 더 컸다(도 49(c) 참조). 이는 A44v11이 시노-PAI-1에 결합할 때 APGv2보다 교환으로부터의 큰 전체적인 보호를 제공할 수 있다는 것을 나타낸다.
비교 연구:
비교 연구를 위해, 3가지의 시노-PAI-1 상태 각각으로부터 생성된 150개의 펩티드 모두에 대한 중수소 흡수를 모니터링하였다. (일반적으로, Wei, et al., Drug Discovery Today, 19(1): 95-102(2014)를 참조). 3가지 상태 각각으로부터의 데이터 도표를 서로 비교하였고, 데이터 해석을 용이하게 하기 위해 나비 도표를 생성하였다(예를 들어, 도 50(a), 51(a), 및 52(a) 참조). 각각의 나비 도표의 경우, x 축은 비교한 150개의 펩티드 각각의 계산된 펩티드 중앙점 위치, i, 이고; y-축은 평균 상대 분획 교환이다(비).
시노-PAI-1 상태 각각의 비교를 위한 회절 도표도 또한 생성하였다(예를 들어, 도 50(b), 51(b), 및 52(b) 참조). 이러한 도표에서, 하나의 상태로부터의 중수소 흡수를 다른 것으로부터 감하고, 나비 도표와 유사하게 도표화한다. 각각의 펩티드에 대한 차의 합은 수직 막대로 나타낸다. 수평 점선은 개별적인 측정(±0.5 Da) 또는 차의 합(±1.1 Da)이 측정의 오차를 초과하는 값을 나타내고, 2가지 상태 사이의 실제 차이인 것으로 간주될 수 있다. 이러한 기술에 관한 추가적인 세부사항은 문헌(Houde D. et al., J. Pharm . Sci. 100(6):2071-86(2011))에 개시되어 있다.
우선, 시노-PAI-1 단독을 A44v11:시노-PAI-1 결합상태에 비교하였다(도 50). 이러한 비교를 위한 나비 도표가 도 50(a)에 나타나 있다. 이러한 비교를 위한 차 도표가 도 50(b)에 나타나 있다. A44v11에 결합한 시노-PAI-1과 유리 형태 시노-PAI-1 사이에서 관찰된 차이는 시노-PAI-1의 2개의 영역 내에 주로 위치한다. 하나의 영역은 N-말단(잔기 44~64)에 가깝고 다른 영역은 C-말단(잔기 307~321)에 가깝다(도 50(b) 참조).
다음으로, 시노-PAI-1 단독을 APGv2:시노-PAI-1 결합 상태에 비교하였다(도 51). 이러한 비교를 위한 나비 도표가 도 51(a)에 나타나 있다. 이러한 비교를 위한 차 도표가 도 51(b)에 나타나 있다. APGv2에 결합된 시노-PAI-1과 유리 형태 시노-PAI-1 사이에서 관찰된 차이는 시노-PAI-1의 2개의 영역 내에 주로 위치한다. 하나의 영역은 N-말단에 가깝고 다른 영역은 C-말단에 가까운데 이는 A44v11:시노-PAI-1 결과와 유사하다. 시노-PAI-1과의 A44v11 및 APGv2 복합체는 결합된 상태에서 감소된 중수소 흡수를 보이는 펩티드를 공유하는데, 이는 2종의 항체에 대한 에피토프가 유사하다는 것을 가리키는 것일 수 있다.
마지막으로, 2종의 항체-결합 시노-PAI-1 상태를 서로 비교하였다(도 52). 이러한 비교를 위한 나비 도표가 도 52(a)에 나타나 있다. 이러한 비교를 위한 차 도표가 도 52(b)에 나타나 있다. A44v11:시노-PAI-1 및 APGv2:시노-PAI-1 사이에서 관찰된 차이는 시노-PAI-1의 C-말단 영역 내에 위치한다. (도 52(b) 참조).
실시예
25: 항체
A44V11
및
APGv2의
에피토프
비교
HDX MS를 A44v11 및 APGv2 항체의 에피토프를 추가로 정의하기 위해 사용하였다. HDX MS에서 생성된 중첩 펩티드를 사용하여, 항체 에피토프를 펩티드-수준 해상도보다 약간 낫도록 개량할 수 있다(예를 들어, 도 48 참조). A44V11 결합으로의 교체에 대한 유의한 보호를 나타내는 펩티드에 대한 HDX MS 데이터를 더 분석하여 시노-PAI-1:A44v11 상호작용에 대한 에피토프를 결정하였다. 시노-PAI-1의 A44V11 에피토프에 대한 HDX 데이터는 결정학 접근법을 사용하여 결정한 에피토프와 일치하는 것으로 밝혀졌다. HDX MS를 사용하여 확인된 시노-PAI-1의 A44V11 에피토프를 도 53(굵은 글씨) 및, 아래에 약칭 형식으로 나타냈다:
T-T-G-G-E-T-R-Q-Q-I-Q(서열 번호 159);
R-H-L (서열 번호 160);
T-D-M-X-X-X-F-Q-A-D-F-T-S-L-S-N-Q-E-P-L-H-V (서열 번호 161)
APGv2 결합으로의 교환으로부터의 유의한 보호를 나타내는 시노-PAI-1 펩티드에 대한 HDX MS 데이터를 분석하여 시노-PAI-1:APGv2 상호작용에 대한 에피토프를 추가로 결정하였다. A44v11 및 APGv2에 대한 HDX MS 에피토프 맵핑 데이터는 도 52에서 일반적으로 나타낸 바와 같이 에피토프가 동일한 영역 내에 있다는 것을 보여준다. 잔기 307~321의 이러한 영역에서, 동일한 펩티드는 A44v11 및 APGv2에 대한 항체 결합 상태에서의 보호를 나타낸다. 그러나, 시노-PAI-1가 APGv2 보다는 A44v11에 결합할 때 보호의 범위가 더 크다(도 49(c) 참조). 이러한 발견은 시노-PAI-1의 잔기 307~321 영역 내 차이 피크를 도시한 도 52(b)에서 더욱 명백해진다. 이는 이러한 시노-PAI-1 및 A44V11 및 APGv2 항체 각각 사이에 만들어진 특이적 접촉 내에 차이가 존재한다는 것을 나타낸다. 따라서, A44V11 및 APGv2의 에피토프가 PAI-1의 유사한 영역 내에 위치하지만, 각각의 항체에 대한 에피토프는 동일하지 않다는 것을 보여준다.
SEQUENCE LISTING
<110> Sanofi
<120> ANTIBODIES TO PLASMINOGEN ACTIVATOR INHIBITOR-1 (PAI-1) AND USES
THEREOF
<130> 13-596-WO
<140> PCT/US2014/050896
<141> 2014-08-13
<160> 170
<170> PatentIn version 3.5
<210> 1
<211> 379
<212> PRT
<213> Homo sapiens
<400> 1
Val His His Pro Pro Ser Tyr Val Ala His Leu Ala Ser Asp Phe Gly
1 5 10 15
Val Arg Val Phe Gln Gln Val Ala Gln Ala Ser Lys Asp Arg Asn Val
20 25 30
Val Phe Ser Pro Tyr Gly Val Ala Ser Val Leu Ala Met Leu Gln Leu
35 40 45
Thr Thr Gly Gly Glu Thr Gln Gln Gln Ile Gln Ala Ala Met Gly Phe
50 55 60
Lys Ile Asp Asp Lys Gly Met Ala Pro Ala Leu Arg His Leu Tyr Lys
65 70 75 80
Glu Leu Met Gly Pro Trp Asn Lys Asp Glu Ile Ser Thr Thr Asp Ala
85 90 95
Ile Phe Val Gln Arg Asp Leu Lys Leu Val Gln Gly Phe Met Pro His
100 105 110
Phe Phe Arg Leu Phe Arg Ser Thr Val Lys Gln Val Asp Phe Ser Glu
115 120 125
Val Glu Arg Ala Arg Phe Ile Ile Asn Asp Trp Val Lys Thr His Thr
130 135 140
Lys Gly Met Ile Ser Asn Leu Leu Gly Lys Gly Ala Val Asp Gln Leu
145 150 155 160
Thr Arg Leu Val Leu Val Asn Ala Leu Tyr Phe Asn Gly Gln Trp Lys
165 170 175
Thr Pro Phe Pro Asp Ser Ser Thr His Arg Arg Leu Phe His Lys Ser
180 185 190
Asp Gly Ser Thr Val Ser Val Pro Met Met Ala Gln Thr Asn Lys Phe
195 200 205
Asn Tyr Thr Glu Phe Thr Thr Pro Asp Gly His Tyr Tyr Asp Ile Leu
210 215 220
Glu Leu Pro Tyr His Gly Asp Thr Leu Ser Met Phe Ile Ala Ala Pro
225 230 235 240
Tyr Glu Lys Glu Val Pro Leu Ser Ala Leu Thr Asn Ile Leu Ser Ala
245 250 255
Gln Leu Ile Ser His Trp Lys Gly Asn Met Thr Arg Leu Pro Arg Leu
260 265 270
Leu Val Leu Pro Lys Phe Ser Leu Glu Thr Glu Val Asp Leu Arg Lys
275 280 285
Pro Leu Glu Asn Leu Gly Met Thr Asp Met Phe Arg Gln Phe Gln Ala
290 295 300
Asp Phe Thr Ser Leu Ser Asp Gln Glu Pro Leu His Val Ala Gln Ala
305 310 315 320
Leu Gln Lys Val Lys Ile Glu Val Asn Glu Ser Gly Thr Val Ala Ser
325 330 335
Ser Ser Thr Ala Val Ile Val Ser Ala Arg Met Ala Pro Glu Glu Ile
340 345 350
Ile Met Asp Arg Pro Phe Leu Phe Val Val Arg His Asn Pro Thr Gly
355 360 365
Thr Val Leu Phe Met Gly Gln Val Met Glu Pro
370 375
<210> 2
<211> 124
<212> PRT
<213> Artificial Sequence
<220>
<223> mA105 VH
<400> 2
Gln Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Met Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Ala Thr Gly Phe Thr Phe Ser Ile Tyr
20 25 30
Trp Ile Glu Trp Val Lys Gln Arg Pro Gly Leu Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Leu Pro Gly Ser Gly Ser Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Lys Ala Thr Phe Thr Ala Asp Thr Ser Ser Asn Thr Ala Phe
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Leu Tyr Tyr Asp Leu Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Ile Leu Thr Val Ser Ser Ala Lys Thr Thr Pro Pro
115 120
<210> 3
<211> 115
<212> PRT
<213> Artificial Sequence
<220>
<223> mA105 VL
<400> 3
Asp Val Val Met Thr Gln Thr Pro Leu Thr Leu Ser Val Thr Ile Gly
1 5 10 15
Gln Pro Ala Ser Ile Ser Cys Lys Ser Ser Gln Ser Leu Leu Asp Ser
20 25 30
Asp Gly Lys Thr Tyr Leu Asn Trp Leu Leu Gln Arg Pro Gly Gln Ser
35 40 45
Pro Gln Arg Leu Ile Ser Leu Val Ser Lys Leu Asp Ser Gly Val Pro
50 55 60
Asp Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Leu
65 70 75 80
Ser Arg Val Glu Gly Ala Asp Leu Gly Val Tyr Tyr Cys Trp Gln Asp
85 90 95
Arg His Phe Pro Arg Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg Ala Asp
115
<210> 4
<211> 125
<212> PRT
<213> Artificial Sequence
<220>
<223> mA39 VH
<400> 4
Gln Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Met Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Ala Thr Gly Tyr Thr Phe Asn Ile Tyr
20 25 30
Trp Ile Gln Trp Val Lys Gln Arg Pro Gly His Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Leu Pro Gly Ser Asn Thr Asn Tyr Asn Glu Lys Phe Lys
50 55 60
Asp Lys Ala Thr Phe Thr Ala Asp Ser Ser Ser Asn Thr Ala Tyr Met
65 70 75 80
Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Leu Gly Ile Gly Leu Arg Gly Ala Leu Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Ser Val Thr Val Ser Ser Ala Lys Thr Thr Pro Pro
115 120 125
<210> 5
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<223> mA39 VL
<400> 5
Asp Ile Gln Met Thr His Ser Pro Ala Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Glu Thr Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Tyr
20 25 30
Leu Ala Trp Tyr His Gln Lys Gln Gly Lys Ser Pro Gln Leu Leu Val
35 40 45
Tyr Asn Ala Lys Thr Leu Ala Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Gln Phe Ser Leu Asn Ile Lys Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Gly Thr Phe Tyr Cys Gln His Arg Tyr Gly Ser Pro Trp
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg Ala Asp
100 105 110
<210> 6
<211> 125
<212> PRT
<213> Artificial Sequence
<220>
<223> mA44 VH
<400> 6
Glu Met Gln Leu Gln Glu Ser Gly Pro Ser Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ser Val Thr Gly Asp Ser Met Thr Asn Gly
20 25 30
Tyr Trp Asn Trp Ile Arg Lys Phe Pro Gly Asn Lys Leu Glu Tyr Met
35 40 45
Gly Tyr Ile Thr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys
50 55 60
Gly Arg Ile Ser Ile Thr Arg Asn Thr Ser Lys Asn Gln Tyr Tyr Leu
65 70 75 80
Gln Leu Ser Ser Val Thr Thr Glu Asp Thr Ala Thr Tyr Tyr Cys Ala
85 90 95
Arg Trp His Tyr Gly Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Leu Thr Val Ser Ser Ala Lys Thr Thr Pro Pro
115 120 125
<210> 7
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<223> mA44 VL
<400> 7
Asp Ile Lys Met Thr Gln Ser Pro Ser Ser Met Tyr Ala Ser Leu Gly
1 5 10 15
Glu Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ser Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Asn Arg Ser Val Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Tyr
65 70 75 80
Glu Asp Met Gly Ile Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg Ala Asp
100 105 110
<210> 8
<211> 124
<212> PRT
<213> Artificial Sequence
<220>
<223> mA71 VH
<400> 8
Gln Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Met Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Ala Thr Gly Phe Thr Phe Ser Thr Tyr
20 25 30
Trp Ile Glu Trp Ile Lys Gln Arg Pro Gly His Gly Leu Asp Trp Ile
35 40 45
Gly Glu Ile Leu Pro Gly Ser Gly Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Lys Ala Thr Phe Thr Ala Asp Thr Ser Ser Asn Thr Val Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Leu Tyr Tyr Asn Leu Asp Ser Trp Gly Gln Gly Thr
100 105 110
Thr Leu Thr Val Ser Ser Ala Lys Thr Thr Pro Pro
115 120
<210> 9
<211> 115
<212> PRT
<213> Artificial Sequence
<220>
<223> mA71 VL
<400> 9
Asp Val Val Met Thr Gln Thr Pro Leu Thr Leu Ser Val Thr Ile Gly
1 5 10 15
Gln Pro Ala Ser Ile Ser Cys Lys Ser Ser Gln Ser Leu Leu Asp Ser
20 25 30
Asp Gly Lys Thr Tyr Leu Tyr Trp Leu Leu Gln Arg Pro Gly Gln Ser
35 40 45
Pro Lys Arg Leu Ile Tyr Leu Val Ser Lys Leu Asp Ser Gly Val Pro
50 55 60
Asp Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Leu Gly Val Tyr Tyr Cys Trp Gln Asp
85 90 95
Thr His Phe Pro Arg Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg Ala Asp
115
<210> 10
<211> 140
<212> PRT
<213> Artificial Sequence
<220>
<223> mB109 VH
<400> 10
Glu Val Gln Leu Gln Gln Ser Gly Ser Val Leu Ala Arg Pro Gly Thr
1 5 10 15
Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Ser Phe Thr Ser Tyr
20 25 30
Trp Met His Trp Val Lys Gln Arg Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ala Ile Tyr Pro Gly Asn Ser Gly Gln Gly Leu Asp Trp Ile Gly
50 55 60
Ala Ile Tyr Pro Gly Asn Ser Asp Thr Thr Tyr Asn Gln Lys Phe Glu
65 70 75 80
Asp Lys Ala Lys Leu Thr Ala Val Ala Ser Ala Ser Thr Ala Tyr Met
85 90 95
Glu Val Ser Ser Leu Thr Asn Glu Asp Ser Ala Val Tyr Tyr Cys Thr
100 105 110
Arg Gly Leu Arg Arg Trp Gly Ala Met Asp Tyr Trp Gly Gln Gly Thr
115 120 125
Ser Val Thr Val Ser Ser Ala Lys Thr Thr Pro Pro
130 135 140
<210> 11
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<223> mB109 VL
<400> 11
Asp Ile Val Met Thr Gln Ser His Lys Phe Met Ser Thr Ser Ala Gly
1 5 10 15
Asp Arg Val Ser Ile Pro Cys Lys Ala Ser Gln Asp Val Ser Ser Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Leu Gly Gln Ser Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Arg Tyr Thr Gly Val Pro Asp Arg Phe Thr Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Val Gln Ala
65 70 75 80
Glu Asp Leu Ala Val Tyr Tyr Cys Gln Gln His Tyr Ser Ser Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Asn Leu Glu Ile Lys Arg Ala Asp
100 105 110
<210> 12
<211> 125
<212> PRT
<213> Artificial Sequence
<220>
<223> mB28 VH
<400> 12
Gln Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Met Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Ala Thr Gly Tyr Thr Phe Ser Ile Ser
20 25 30
Trp Ile Glu Trp Ile Lys Gln Arg Pro Gly Gly Leu Glu Trp Ile Gly
35 40 45
Lys Ile Leu Pro Gly Ser Gly Gly Ala Asn Tyr Asn Glu Lys Phe Lys
50 55 60
Gly Lys Ala Thr Val Thr Ala Asp Thr Ser Ser Asn Thr Val Tyr Met
65 70 75 80
Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Leu Ser Thr Gly Thr Arg Gly Ala Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Leu Thr Val Ser Ser Ala Lys Thr Thr Pro Pro
115 120 125
<210> 13
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<223> mB28 VL
<400> 13
Asp Ile Gln Leu Thr Gln Ser Pro Ala Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Ala Thr Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Val Tyr Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Gln Gly Lys Ser Pro Gln Leu Leu Val
35 40 45
Tyr Asn Ala Lys Thr Leu Ala Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Gln Phe Ser Leu Lys Ile Asn Tyr Leu Gln Pro
65 70 75 80
Glu Asp Phe Gly Ser Tyr Tyr Cys Gln His His Tyr Gly Thr Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Ala Asp
100 105 110
<210> 14
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> mC45 VH
<400> 14
Gln Val Gln Leu Gln Gln Ser Gly Val Glu Leu Val Arg Pro Gly Thr
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Ala Phe Thr Asn Tyr
20 25 30
Leu Ile Glu Trp Ile Lys Gln Arg Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile His Pro Gly Ser Gly Val Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Lys Ala Ile Leu Thr Ala Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Asp Asp Ser Ala Val Tyr Phe Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser His Gly Leu Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Ser Val Thr Val Ser Ser
115 120
<210> 15
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> mC45 VL
<400> 15
Asp Ile Lys Met Thr Gln Ser Pro Ser Ser Met Tyr Ala Ser Leu Gly
1 5 10 15
Glu Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Phe Gln Gln Lys Pro Gly Lys Ser Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Asn Arg Leu Val Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Tyr
65 70 75 80
Glu Asp Met Gly Ile Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 16
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> mE16 VH
<400> 16
Glu Val Lys Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Thr Pro Glu Lys Gly Leu Gly Trp Val
35 40 45
Ala Ser Leu Arg Thr Gly Gly Asn Thr Tyr Tyr Ser Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Asp Arg Asn Ile Leu Tyr Leu
65 70 75 80
Gln Met Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Leu Arg His Trp Gly Tyr Phe Asp Val Trp Gly Ala Gly Thr
100 105 110
Thr Val Thr Val Ser Ser
115
<210> 17
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> mE16 VL
<400> 17
Asp Ile Val Met Thr Gln Ser His Lys Phe Met Ser Thr Ser Val Gly
1 5 10 15
Asp Arg Val Asn Ile Thr Cys Lys Ala Ser Gln Asp Val Ser Thr Ala
20 25 30
Val Gly Trp Tyr Gln Gln Glu Pro Gly Gln Ser Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Asn Arg His Thr Gly Val Pro Asp Arg Phe Thr Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Val Gln Ala
65 70 75 80
Glu Asp Leu Ala Val Tyr Tyr Cys Gln Gln His Tyr Ser Ser Pro Trp
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 18
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> mE21 VH
<400> 18
Glu Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Val Arg Ser Gly Ala
1 5 10 15
Ser Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Tyr
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Asp Pro Glu Asn Gly Asp Thr Glu Tyr Asp Pro Lys Phe
50 55 60
Gln Ala Lys Ala Thr Met Thr Ala Asp Thr Ser Ser Asn Thr Ala Tyr
65 70 75 80
Leu Gln Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Met Tyr Gly Asn Tyr Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Thr Leu Thr Val Ser Ser
115
<210> 19
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> mE21 VL
<400> 19
Asp Ile Gln Met Thr Gln Thr Thr Ser Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Asp Arg Val Thr Ile Ser Cys Arg Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Asp Gly Thr Val Lys Leu Leu Ile
35 40 45
Tyr Tyr Thr Ser Arg Leu His Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Ser Leu Thr Ile Ser Asn Leu Glu Gln
65 70 75 80
Glu Asp Ile Ala Thr Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Trp
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 20
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> mA105 HCDR3
<400> 20
Ala Arg Gly Gly Leu Tyr Tyr Asp Leu Asp Tyr
1 5 10
<210> 21
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> mA105 HCDR2
<400> 21
Ile Leu Pro Gly Ser Gly Ser Thr
1 5
<210> 22
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> mA105 HCDR1
<400> 22
Gly Phe Thr Phe Ser Ile Tyr Trp
1 5
<210> 23
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> mA105 LCDR3
<400> 23
Trp Gln Asp Arg His Phe Pro Arg Thr
1 5
<210> 24
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> mA105 LCDR2
<400> 24
Leu Val Ser
1
<210> 25
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> mA105 LCDR1
<400> 25
Gln Ser Leu Leu Asp Ser Asp Gly Lys Thr Tyr
1 5 10
<210> 26
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> mA39 HCDR3
<400> 26
Ala Arg Leu Gly Ile Gly Leu Arg Gly Ala Leu Asp Tyr
1 5 10
<210> 27
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> mA39 HCDR2
<400> 27
Ile Leu Pro Gly Ser Asn Thr
1 5
<210> 28
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> mA39 HCDR1
<400> 28
Gly Tyr Thr Phe Asn Ile Tyr Trp
1 5
<210> 29
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> mA39 LCDR3
<400> 29
Gln His Arg Tyr Gly Ser Pro Trp Thr
1 5
<210> 30
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> mA39 LCDR2
<400> 30
Asn Ala Lys
1
<210> 31
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> mA38 LCDR1
<400> 31
Glu Asn Ile Tyr Ser Tyr
1 5
<210> 32
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> mA44 HCDR3
<400> 32
Ala Arg Trp His Tyr Gly Ser Pro Tyr Tyr Phe Asp Tyr
1 5 10
<210> 33
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> mA44 HCDR2
<400> 33
Ile Thr Tyr Ser Gly Ser Thr
1 5
<210> 34
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> mA44 HCDR1
<400> 34
Gly Asp Ser Met Thr Asn Gly Tyr
1 5
<210> 35
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> mA44 LCDR3
<400> 35
Leu Gln Tyr Asp Glu Phe Pro Pro Thr
1 5
<210> 36
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> mA44 LCDR2
<400> 36
Arg Ala Asn
1
<210> 37
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> mA44 LCDR1
<400> 37
Gln Asp Ile Asn Ser Tyr
1 5
<210> 38
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> mA71 HCDR3
<400> 38
Ala Arg Gly Gly Leu Tyr Tyr Asn Leu Asp Ser
1 5 10
<210> 39
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> mA71 HCDR2
<400> 39
Ile Leu Pro Gly Ser Gly Asn Thr
1 5
<210> 40
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> mA71 HCDR1
<400> 40
Gly Phe Thr Phe Ser Thr Tyr Trp
1 5
<210> 41
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> mA71 LCDR3
<400> 41
Trp Gln Asp Thr His Phe Pro Arg Thr
1 5
<210> 42
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> mA71 LCDR2
<400> 42
Leu Val Ser
1
<210> 43
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> mA71 LCDR1
<400> 43
Gln Ser Leu Leu Asp Ser Asp Gly Lys Thr Tyr
1 5 10
<210> 44
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> mA75 HCDR3
<400> 44
Ala Arg Gly Gly Leu Tyr Tyr Ala Met Asp Tyr
1 5 10
<210> 45
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> mA75 HCDR2
<400> 45
Ile Leu Pro Gly Ser Gly Leu Thr
1 5
<210> 46
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> mA75 HCDR1
<400> 46
Gly Phe Thr Phe Ser Thr Tyr Trp
1 5
<210> 47
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> mA75 LCDR3
<400> 47
Trp Gln Gly Ser His Phe Pro Gln Thr
1 5
<210> 48
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> mA75 LCDR2
<400> 48
Leu Val Cys
1
<210> 49
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> mA75 LCDR1
<400> 49
Gln Ser Leu Leu Asp Ser Glu Gly Lys Thr Tyr
1 5 10
<210> 50
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> mB109 HCDR3
<400> 50
Thr Arg Gly Leu Arg Arg Trp Gly Ala Met Asp Tyr
1 5 10
<210> 51
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> mB109 HCDR2
<400> 51
Ile Leu Pro Gly Ser Gly Leu Thr
1 5
<210> 52
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> mB109 HCDR1
<400> 52
Gly Phe Thr Phe Ser Thr Tyr Trp
1 5
<210> 53
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> mB109 LCDR3
<400> 53
Gln Gln His Tyr Ser Ser Pro Tyr Thr
1 5
<210> 54
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> mB109 LCDR2
<400> 54
Ser Ala Ser
1
<210> 55
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> mB109 LCDR1
<400> 55
Gln Asp Val Ser Ser Ala
1 5
<210> 56
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> mB28 HCDR3
<400> 56
Ala Arg Leu Ser Thr Gly Thr Arg Gly Ala Phe Asp Tyr
1 5 10
<210> 57
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> mB28 HCDR2
<400> 57
Ile Leu Pro Gly Ser Gly Gly Ala
1 5
<210> 58
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> mB28 HCDR1
<400> 58
Gly Tyr Thr Phe Ser Ile Ser Trp
1 5
<210> 59
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> mB28 LCDR3
<400> 59
Gln His His Tyr Gly Thr Pro Pro Thr
1 5
<210> 60
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> mB28 LCDR2
<400> 60
Asn Ala Lys
1
<210> 61
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> mB28 LCDR1
<400> 61
Glu Asn Val Tyr Ser Tyr
1 5
<210> 62
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> mC45 HCDR3
<400> 62
Ala Arg Asp Tyr Tyr Gly Ser Ser His Gly Leu Met Asp Tyr
1 5 10
<210> 63
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> mC45 HCDR2
<400> 63
Ile His Pro Gly Ser Gly Val Thr
1 5
<210> 64
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> mC45 HCDR1
<400> 64
Gly Tyr Ala Phe Thr Asn Tyr Leu
1 5
<210> 65
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> mC45 LCDR3
<400> 65
Leu Gln Tyr Asp Glu Phe Pro Arg Thr
1 5
<210> 66
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> mC45 LCDR2
<400> 66
Arg Ala Asn
1
<210> 67
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> mC45 LCDR1
<400> 67
Gln Asp Ile Asn Ser Tyr
1 5
<210> 68
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> mE16 HCDR3
<400> 68
Ala Arg Gly Leu Arg His Trp Gly Tyr Phe Asp Val
1 5 10
<210> 69
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> mE16 HCDR2
<400> 69
Leu Arg Thr Gly Gly Asn Thr
1 5
<210> 70
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> mE16 HCDR1
<400> 70
Gly Phe Thr Phe Ser Asn Tyr Gly
1 5
<210> 71
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> mE16 LCDR3
<400> 71
Gln Gln His Tyr Ser Ser Pro Trp Thr
1 5
<210> 72
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> mE16 LCDR2
<400> 72
Ser Ala Ser
1
<210> 73
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> mE16 LCDR1
<400> 73
Gln Asp Ile Ser Asn Tyr
1 5
<210> 74
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> mE21 HCDR3
<400> 74
Met Tyr Gly Asn Tyr Pro Tyr Tyr Phe Asp Tyr
1 5 10
<210> 75
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> mE21 HCDR2
<400> 75
Ile Asp Pro Glu Asn Gly Asp Thr
1 5
<210> 76
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> mE21 HCDR1
<400> 76
Gly Phe Asn Ile Lys Asp Tyr Tyr
1 5
<210> 77
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> mE21 LCDR3
<400> 77
Gln Gln Gly Asn Thr Leu Pro Trp Thr
1 5
<210> 78
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> mE21 LCDR2
<400> 78
Tyr Thr Ser
1
<210> 79
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> mE21 LCDR1
<400> 79
Gln Asp Ile Ser Asn Tyr
1 5
<210> 80
<211> 124
<212> PRT
<213> Artificial Sequence
<220>
<223> mA75 VH
<400> 80
Gln Gly Gln Leu Gln Gln Ser Gly Ala Glu Leu Met Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Phe Thr Phe Ser Thr Tyr
20 25 30
Trp Ile Ala Trp Leu Lys Gln Arg Pro Gly His Gly Leu Glu Trp Ile
35 40 45
Ala Glu Ile Leu Pro Gly Ser Gly Leu Thr Asn Tyr Asn Glu Ile Phe
50 55 60
Arg Gly Lys Ala Thr Phe Thr Ala Asp Thr Ser Ser Asn Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Leu Tyr Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Ser Val Thr Val Ser Ser Ala Lys Thr Thr Ala Pro
115 120
<210> 81
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<223> mA75 VL
<400> 81
Asp Val Val Met Thr Gln Thr Pro Leu Thr Leu Ser Val Thr Ile Gly
1 5 10 15
Gln Pro Ala Ser Ile Cys Lys Ser Ser Gln Ser Leu Leu Asp Ser Glu
20 25 30
Gly Lys Thr Tyr Leu Asn Trp Leu Phe Gln Arg Pro Gly Gln Ser Pro
35 40 45
Lys Arg Leu Ile Tyr Leu Val Cys Lys Leu Asp Cys Gly Val Pro Asp
50 55 60
Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile Ser
65 70 75 80
Arg Val Glu Gly Glu Asp Leu Gly Val Tyr Tyr Cys Trp Gln Gly Ser
85 90 95
His Phe Pro Gln Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg
100 105 110
Ala Asp
<210> 82
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> HC1a
<400> 82
Glu Met Thr Leu Lys Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ser Val Thr Gly Asp Ser Met Thr Asn Gly
20 25 30
Tyr Trp Asn Trp Ile Arg Lys Phe Pro Gly Lys Ala Leu Glu Tyr Met
35 40 45
Gly Tyr Ile Thr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys
50 55 60
Gly Arg Ile Ser Ile Thr Arg Asn Thr Ser Lys Asn Gln Tyr Tyr Leu
65 70 75 80
Thr Leu Ser Ser Val Thr Thr Val Asp Thr Ala Thr Tyr Tyr Cys Ala
85 90 95
Arg Trp His Tyr Gly Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Leu Thr Val Ser Ser
115
<210> 83
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> HC1b
<400> 83
Glu Met Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Ser Val Thr Gly Asp Ser Met Thr Asn Gly
20 25 30
Tyr Trp Asn Trp Ile Arg Lys Phe Pro Gly Lys Gly Leu Glu Tyr Met
35 40 45
Gly Tyr Ile Thr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys
50 55 60
Gly Arg Ile Ser Ile Thr Arg Asn Thr Ser Lys Asn Gln Tyr Tyr Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Thr Ala Asp Thr Ala Thr Tyr Tyr Cys Ala
85 90 95
Arg Trp His Tyr Gly Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Leu Thr Val Ser Ser
115
<210> 84
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> HC2a
<400> 84
Glu Met Thr Leu Lys Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ser Val Thr Gly Glu Ser Met Thr Gln Gly
20 25 30
Tyr Trp Asn Trp Ile Arg Lys Phe Pro Gly Lys Ala Leu Glu Tyr Met
35 40 45
Gly Tyr Ile Thr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys
50 55 60
Gly Arg Ile Ser Ile Thr Arg Gln Thr Ser Lys Asn Gln Tyr Tyr Leu
65 70 75 80
Thr Leu Ser Ser Val Thr Thr Val Glu Thr Ala Thr Tyr Tyr Cys Ala
85 90 95
Arg Trp His Tyr Gly Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Leu Thr Val Ser Ser
115
<210> 85
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> HC2b
<400> 85
Glu Met Thr Leu Lys Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ser Val Thr Gly Asp Ser Met Thr Gln Gly
20 25 30
Tyr Trp Asn Trp Ile Arg Lys Phe Pro Gly Lys Ala Leu Glu Tyr Met
35 40 45
Gly Tyr Ile Thr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys
50 55 60
Gly Arg Ile Ser Ile Thr Arg Asn Thr Ser Lys Asn Gln Tyr Tyr Leu
65 70 75 80
Thr Leu Ser Ser Val Thr Thr Val Asp Thr Ala Thr Tyr Tyr Cys Ala
85 90 95
Arg Trp His Tyr Gly Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Leu Thr Val Ser Ser
115
<210> 86
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> HC3
<400> 86
Gln Met Thr Leu Lys Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ser Val Ser Gly Asp Ser Met Thr Asn Gly
20 25 30
Tyr Trp Asn Trp Ile Arg Gln Phe Pro Gly Lys Ala Leu Glu Tyr Met
35 40 45
Gly Tyr Ile Thr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys
50 55 60
Gly Arg Ile Thr Ile Thr Arg Asp Thr Ser Lys Asn Gln Tyr Tyr Leu
65 70 75 80
Thr Leu Ser Ser Val Thr Thr Val Asp Thr Ala Thr Tyr Tyr Cys Ala
85 90 95
Arg Trp His Tyr Gly Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Leu Thr Val Ser Ser
115
<210> 87
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> HC4
<400> 87
Gln Met Thr Leu Lys Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ser Val Ser Gly Glu Ser Met Thr Gln Gly
20 25 30
Tyr Trp Asn Trp Ile Arg Gln Phe Pro Gly Lys Ala Leu Glu Tyr Met
35 40 45
Gly Tyr Ile Thr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys
50 55 60
Gly Arg Ile Thr Ile Thr Arg Gln Thr Ser Lys Asn Gln Tyr Tyr Leu
65 70 75 80
Thr Leu Ser Ser Val Thr Thr Val Glu Thr Ala Thr Tyr Tyr Cys Ala
85 90 95
Arg Trp His Tyr Gly Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Leu Thr Val Ser Ser
115
<210> 88
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> HC5a
<400> 88
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Asp Ser Met Thr Asn Gly
20 25 30
Tyr Trp Asn Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Tyr Met
35 40 45
Gly Tyr Ile Thr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Ile Thr Ile Ser Arg Asn Thr Ser Lys Asn Gln Tyr Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Trp His Tyr Gly Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 89
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> HC5b
<400> 89
Gln Met Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Asp Ser Met Thr Asn Gly
20 25 30
Tyr Trp Asn Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Tyr Met
35 40 45
Gly Tyr Ile Thr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Ile Thr Ile Ser Arg Asp Thr Ser Lys Asn Gln Tyr Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Trp His Tyr Gly Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 90
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> HC5c
<400> 90
Gln Met Gln Leu Gln Gln Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Ile Ser Gly Asp Ser Met Thr Asn Gly
20 25 30
Tyr Trp Asn Trp Ile Arg Gln Ser Pro Ser Arg Gly Leu Glu Tyr Met
35 40 45
Gly Tyr Ile Thr Tyr Ser Gly Ser Thr Tyr Tyr Ala Val Ser Val Lys
50 55 60
Ser Arg Ile Thr Ile Asn Arg Asp Thr Ser Lys Asn Gln Tyr Ser Leu
65 70 75 80
Gln Leu Ser Ser Val Thr Pro Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Trp His Tyr Gly Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 91
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> LC1a
<400> 91
Asp Ile Lys Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ser Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Asn Arg Ser Val Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Ser Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Leu Gly Ile Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 92
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> LC1b
<400> 92
Asp Ile Lys Met Thr Gln Ser Pro Ser Ser Val Ser Val Ser Pro Gly
1 5 10 15
Gln Thr Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Gln Ser Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Asn Arg Ser Val Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Ser Leu Thr Ile Ser Ser Leu Gln Ala
65 70 75 80
Met Asp Glu Gly Ile Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Thr Ile Lys
100 105
<210> 93
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> LC2
<400> 93
Asp Ile Lys Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ser Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Gln Arg Ser Val Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Ser Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Leu Gly Ile Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 94
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> LC3
<400> 94
Asp Ile Lys Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ser Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Asn Arg Ser Val Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Ser Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Leu Ala Thr Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 95
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> LC4
<400> 95
Asp Ile Lys Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ser Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Gln Arg Ser Val Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Ser Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Leu Ala Thr Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 96
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> LC5a
<400> 96
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Asn Arg Ser Val Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 97
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> LC5b
<400> 97
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ala Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Asn Arg Ser Val Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 98
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> LC5c
<400> 98
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Gln Ala Pro Arg Thr Leu Ile
35 40 45
Tyr Arg Ala Asn Arg Ser Val Asp Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 99
<211> 255
<212> PRT
<213> Artificial Sequence
<220>
<223> CH
<400> 99
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
225 230 235 240
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
245 250 255
<210> 100
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> CL
<400> 100
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
1 5 10 15
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
20 25 30
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
35 40 45
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
50 55 60
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
65 70 75 80
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
85 90 95
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
100 105
<210> 101
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> vk1
<400> 101
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Leu Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 102
<211> 106
<212> PRT
<213> Artificial Sequence
<220>
<223> vlambda3
<220>
<221> misc_feature
<222> (22)..(22)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (87)..(87)
<223> Xaa can be any naturally occurring amino acid
<400> 102
Ser Tyr Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Ser Ile Thr Xaa Ser Gly Asp Lys Leu Gly Asp Lys Tyr Ala
20 25 30
Ser Trp Tyr Gln Gln Lys Pro Gly Gln Ser Pro Val Leu Val Ile Tyr
35 40 45
Gln Asp Ser Lys Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Met
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Xaa Gln Ala Trp Asp Ser Ser Ala Val Val
85 90 95
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 103
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> vh2
<400> 103
Gln Val Thr Leu Lys Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Phe Ser Gly Phe Ser Leu Ser Thr Ser
20 25 30
Gly Val Gly Val Gly Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala Arg Ile Asp Trp Asp Asp Asp Lys Tyr Tyr Ser Thr Ser
50 55 60
Leu Lys Thr Arg Leu Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Met Gly Phe Thr Gly Thr Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 104
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> vh4
<220>
<221> misc_feature
<222> (22)..(22)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (95)..(95)
<223> Xaa can be any naturally occurring amino acid
<400> 104
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Xaa Thr Val Ser Gly Gly Ser Ile Ser Ser Tyr
20 25 30
Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Asn Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Xaa Ala
85 90 95
Arg Gly Asp Ser Ser Gly Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 105
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Heavy Chain 3'- Primer
<400> 105
tatgcaaggc ttacaaccac a 21
<210> 106
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Light Chain 3'- Primer
<400> 106
ctcattcctg ttgaagctct tgag 24
<210> 107
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> IGKV1-33-01_IGKJ4-01
<400> 107
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 108
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> IGHV4-59-02_IGHD6-13-01_IGHJ4-02
<400> 108
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Val Ser Ser Tyr
20 25 30
Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Asn Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Tyr Ser Ser Ser Trp Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 109
<211> 226
<212> PRT
<213> Artificial Sequence
<220>
<223> A44-hv1LC1a x HC1a
<400> 109
Asp Ile Lys Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ser Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Asn Arg Ser Val Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Ser Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Leu Gly Ile Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Glu Met Thr Leu Lys
100 105 110
Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln Thr Leu Ser Leu Thr
115 120 125
Cys Ser Val Thr Gly Asp Ser Met Thr Asn Gly Tyr Trp Asn Trp Ile
130 135 140
Arg Lys Phe Pro Gly Lys Ala Leu Glu Tyr Met Gly Tyr Ile Thr Tyr
145 150 155 160
Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys Gly Arg Ile Ser Ile
165 170 175
Thr Arg Asn Thr Ser Lys Asn Gln Tyr Tyr Leu Thr Leu Ser Ser Val
180 185 190
Thr Thr Val Asp Thr Ala Thr Tyr Tyr Cys Ala Arg Trp His Tyr Gly
195 200 205
Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val
210 215 220
Ser Ser
225
<210> 110
<211> 226
<212> PRT
<213> Artificial Sequence
<220>
<223> A44-hv2LC1b x HC1b
<400> 110
Asp Ile Lys Met Thr Gln Ser Pro Ser Ser Val Ser Val Ser Pro Gly
1 5 10 15
Gln Thr Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Gln Ser Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Asn Arg Ser Val Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Ser Leu Thr Ile Ser Ser Leu Gln Ala
65 70 75 80
Met Asp Glu Gly Ile Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Thr Ile Lys Glu Met Gln Leu Gln
100 105 110
Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr Leu Ser Leu Thr
115 120 125
Cys Ser Val Thr Gly Asp Ser Met Thr Asn Gly Tyr Trp Asn Trp Ile
130 135 140
Arg Lys Phe Pro Gly Lys Gly Leu Glu Tyr Met Gly Tyr Ile Thr Tyr
145 150 155 160
Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys Gly Arg Ile Ser Ile
165 170 175
Thr Arg Asn Thr Ser Lys Asn Gln Tyr Tyr Leu Lys Leu Ser Ser Val
180 185 190
Thr Thr Ala Asp Thr Ala Thr Tyr Tyr Cys Ala Arg Trp His Tyr Gly
195 200 205
Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val
210 215 220
Ser Ser
225
<210> 111
<211> 226
<212> PRT
<213> Artificial Sequence
<220>
<223> A44-hv3LC2 x HC2a
<400> 111
Asp Ile Lys Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ser Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Gln Arg Ser Val Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Ser Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Leu Gly Ile Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Glu Met Thr Leu Lys
100 105 110
Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln Thr Leu Ser Leu Thr
115 120 125
Cys Ser Val Thr Gly Glu Ser Met Thr Gln Gly Tyr Trp Asn Trp Ile
130 135 140
Arg Lys Phe Pro Gly Lys Ala Leu Glu Tyr Met Gly Tyr Ile Thr Tyr
145 150 155 160
Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys Gly Arg Ile Ser Ile
165 170 175
Thr Arg Gln Thr Ser Lys Asn Gln Tyr Tyr Leu Thr Leu Ser Ser Val
180 185 190
Thr Thr Val Glu Thr Ala Thr Tyr Tyr Cys Ala Arg Trp His Tyr Gly
195 200 205
Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val
210 215 220
Ser Ser
225
<210> 112
<211> 226
<212> PRT
<213> Artificial Sequence
<220>
<223> A44-hv4LC1a x HC2b
<400> 112
Asp Ile Lys Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ser Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Asn Arg Ser Val Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Ser Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Leu Gly Ile Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Glu Met Thr Leu Lys
100 105 110
Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln Thr Leu Ser Leu Thr
115 120 125
Cys Ser Val Thr Gly Asp Ser Met Thr Gln Gly Tyr Trp Asn Trp Ile
130 135 140
Arg Lys Phe Pro Gly Lys Ala Leu Glu Tyr Met Gly Tyr Ile Thr Tyr
145 150 155 160
Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys Gly Arg Ile Ser Ile
165 170 175
Thr Arg Asn Thr Ser Lys Asn Gln Tyr Tyr Leu Thr Leu Ser Ser Val
180 185 190
Thr Thr Val Asp Thr Ala Thr Tyr Tyr Cys Ala Arg Trp His Tyr Gly
195 200 205
Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val
210 215 220
Ser Ser
225
<210> 113
<211> 226
<212> PRT
<213> Artificial Sequence
<220>
<223> A44-hv5LC2 x HC2b
<400> 113
Asp Ile Lys Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ser Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Gln Arg Ser Val Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Ser Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Leu Gly Ile Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Glu Met Thr Leu Lys
100 105 110
Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln Thr Leu Ser Leu Thr
115 120 125
Cys Ser Val Thr Gly Asp Ser Met Thr Gln Gly Tyr Trp Asn Trp Ile
130 135 140
Arg Lys Phe Pro Gly Lys Ala Leu Glu Tyr Met Gly Tyr Ile Thr Tyr
145 150 155 160
Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys Gly Arg Ile Ser Ile
165 170 175
Thr Arg Asn Thr Ser Lys Asn Gln Tyr Tyr Leu Thr Leu Ser Ser Val
180 185 190
Thr Thr Val Asp Thr Ala Thr Tyr Tyr Cys Ala Arg Trp His Tyr Gly
195 200 205
Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val
210 215 220
Ser Ser
225
<210> 114
<211> 226
<212> PRT
<213> Artificial Sequence
<220>
<223> A44-hv6LC3 x HC3
<400> 114
Asp Ile Lys Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ser Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Asn Arg Ser Val Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Ser Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Leu Ala Thr Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Gln Met Thr Leu Lys
100 105 110
Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln Thr Leu Ser Leu Thr
115 120 125
Cys Ser Val Ser Gly Asp Ser Met Thr Asn Gly Tyr Trp Asn Trp Ile
130 135 140
Arg Gln Phe Pro Gly Lys Ala Leu Glu Tyr Met Gly Tyr Ile Thr Tyr
145 150 155 160
Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys Gly Arg Ile Thr Ile
165 170 175
Thr Arg Asp Thr Ser Lys Asn Gln Tyr Tyr Leu Thr Leu Ser Ser Val
180 185 190
Thr Thr Val Asp Thr Ala Thr Tyr Tyr Cys Ala Arg Trp His Tyr Gly
195 200 205
Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val
210 215 220
Ser Ser
225
<210> 115
<211> 226
<212> PRT
<213> Artificial Sequence
<220>
<223> A44-hv7LC4 x HC4
<400> 115
Asp Ile Lys Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ser Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Gln Arg Ser Val Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Ser Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Leu Ala Thr Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Gln Met Thr Leu Lys
100 105 110
Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln Thr Leu Ser Leu Thr
115 120 125
Cys Ser Val Ser Gly Glu Ser Met Thr Gln Gly Tyr Trp Asn Trp Ile
130 135 140
Arg Gln Phe Pro Gly Lys Ala Leu Glu Tyr Met Gly Tyr Ile Thr Tyr
145 150 155 160
Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys Gly Arg Ile Thr Ile
165 170 175
Thr Arg Gln Thr Ser Lys Asn Gln Tyr Tyr Leu Thr Leu Ser Ser Val
180 185 190
Thr Thr Val Glu Thr Ala Thr Tyr Tyr Cys Ala Arg Trp His Tyr Gly
195 200 205
Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val
210 215 220
Ser Ser
225
<210> 116
<211> 226
<212> PRT
<213> Artificial Sequence
<220>
<223> A44-hv8LC5a x HC5a
<400> 116
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Asn Arg Ser Val Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Gln Val Gln Leu Gln
100 105 110
Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr Leu Ser Leu Thr
115 120 125
Cys Thr Val Ser Gly Asp Ser Met Thr Asn Gly Tyr Trp Asn Trp Ile
130 135 140
Arg Gln Pro Pro Gly Lys Gly Leu Glu Tyr Met Gly Tyr Ile Thr Tyr
145 150 155 160
Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys Ser Arg Ile Thr Ile
165 170 175
Ser Arg Asn Thr Ser Lys Asn Gln Tyr Ser Leu Lys Leu Ser Ser Val
180 185 190
Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala Arg Trp His Tyr Gly
195 200 205
Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
210 215 220
Ser Ser
225
<210> 117
<211> 226
<212> PRT
<213> Artificial Sequence
<220>
<223> A44-hv9LC5b x HC5b
<400> 117
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ala Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Asn Arg Ser Val Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Gln Met Gln Leu Gln
100 105 110
Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr Leu Ser Leu Thr
115 120 125
Cys Thr Val Ser Gly Asp Ser Met Thr Asn Gly Tyr Trp Asn Trp Ile
130 135 140
Arg Gln Pro Pro Gly Lys Gly Leu Glu Tyr Met Gly Tyr Ile Thr Tyr
145 150 155 160
Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys Ser Arg Ile Thr Ile
165 170 175
Ser Arg Asp Thr Ser Lys Asn Gln Tyr Ser Leu Lys Leu Ser Ser Val
180 185 190
Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala Arg Trp His Tyr Gly
195 200 205
Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
210 215 220
Ser Ser
225
<210> 118
<211> 226
<212> PRT
<213> Artificial Sequence
<220>
<223> A44-hv10LC5c x HC5c
<400> 118
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Gln Ala Pro Arg Thr Leu Ile
35 40 45
Tyr Arg Ala Asn Arg Ser Val Asp Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Gln Met Gln Leu Gln
100 105 110
Gln Ser Gly Pro Gly Leu Val Lys Pro Ser Gln Thr Leu Ser Leu Thr
115 120 125
Cys Ala Ile Ser Gly Asp Ser Met Thr Asn Gly Tyr Trp Asn Trp Ile
130 135 140
Arg Gln Ser Pro Ser Arg Gly Leu Glu Tyr Met Gly Tyr Ile Thr Tyr
145 150 155 160
Ser Gly Ser Thr Tyr Tyr Ala Val Ser Val Lys Ser Arg Ile Thr Ile
165 170 175
Asn Arg Asp Thr Ser Lys Asn Gln Tyr Ser Leu Gln Leu Ser Ser Val
180 185 190
Thr Pro Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg Trp His Tyr Gly
195 200 205
Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
210 215 220
Ser Ser
225
<210> 119
<211> 226
<212> PRT
<213> Artificial Sequence
<220>
<223> A44-hv11LC2 x HC3
<400> 119
Asp Ile Lys Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ser Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Gln Arg Ser Val Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Ser Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Leu Gly Ile Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Gln Met Thr Leu Lys
100 105 110
Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln Thr Leu Ser Leu Thr
115 120 125
Cys Ser Val Ser Gly Asp Ser Met Thr Asn Gly Tyr Trp Asn Trp Ile
130 135 140
Arg Gln Phe Pro Gly Lys Ala Leu Glu Tyr Met Gly Tyr Ile Thr Tyr
145 150 155 160
Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys Gly Arg Ile Thr Ile
165 170 175
Thr Arg Asp Thr Ser Lys Asn Gln Tyr Tyr Leu Thr Leu Ser Ser Val
180 185 190
Thr Thr Val Asp Thr Ala Thr Tyr Tyr Cys Ala Arg Trp His Tyr Gly
195 200 205
Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val
210 215 220
Ser Ser
225
<210> 120
<211> 226
<212> PRT
<213> Artificial Sequence
<220>
<223> A44-hv12LC4 X HC3
<400> 120
Asp Ile Lys Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ser Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Gln Arg Ser Val Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Ser Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Leu Ala Thr Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Gln Met Thr Leu Lys
100 105 110
Glu Ser Gly Pro Thr Leu Val Lys Pro Thr Gln Thr Leu Ser Leu Thr
115 120 125
Cys Ser Val Ser Gly Asp Ser Met Thr Asn Gly Tyr Trp Asn Trp Ile
130 135 140
Arg Gln Phe Pro Gly Lys Ala Leu Glu Tyr Met Gly Tyr Ile Thr Tyr
145 150 155 160
Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys Gly Arg Ile Thr Ile
165 170 175
Thr Arg Asp Thr Ser Lys Asn Gln Tyr Tyr Leu Thr Leu Ser Ser Val
180 185 190
Thr Thr Val Asp Thr Ala Thr Tyr Tyr Cys Ala Arg Trp His Tyr Gly
195 200 205
Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val
210 215 220
Ser Ser
225
<210> 121
<211> 226
<212> PRT
<213> Artificial Sequence
<220>
<223> A44-hv13LC2 x HC5b
<400> 121
Asp Ile Lys Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ser Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Gln Arg Ser Val Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Ser Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Leu Gly Ile Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Gln Met Gln Leu Gln
100 105 110
Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr Leu Ser Leu Thr
115 120 125
Cys Thr Val Ser Gly Asp Ser Met Thr Asn Gly Tyr Trp Asn Trp Ile
130 135 140
Arg Gln Pro Pro Gly Lys Gly Leu Glu Tyr Met Gly Tyr Ile Thr Tyr
145 150 155 160
Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys Ser Arg Ile Thr Ile
165 170 175
Ser Arg Asp Thr Ser Lys Asn Gln Tyr Ser Leu Lys Leu Ser Ser Val
180 185 190
Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala Arg Trp His Tyr Gly
195 200 205
Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
210 215 220
Ser Ser
225
<210> 122
<211> 226
<212> PRT
<213> Artificial Sequence
<220>
<223> A44-hv14LC4 x HC5b
<400> 122
Asp Ile Lys Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ser Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Gln Arg Ser Val Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Ser Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Leu Ala Thr Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Gln Met Gln Leu Gln
100 105 110
Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr Leu Ser Leu Thr
115 120 125
Cys Thr Val Ser Gly Asp Ser Met Thr Asn Gly Tyr Trp Asn Trp Ile
130 135 140
Arg Gln Pro Pro Gly Lys Gly Leu Glu Tyr Met Gly Tyr Ile Thr Tyr
145 150 155 160
Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys Ser Arg Ile Thr Ile
165 170 175
Ser Arg Asp Thr Ser Lys Asn Gln Tyr Ser Leu Lys Leu Ser Ser Val
180 185 190
Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala Arg Trp His Tyr Gly
195 200 205
Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
210 215 220
Ser Ser
225
<210> 123
<211> 255
<212> DNA
<213> Artificial Sequence
<220>
<223> HC1a
<400> 123
gagatgaccc tgaaagagtc cggccccacc ctggtcaaac ccacccagac cctgagcctg 60
acctgcagcg tgaccggcga cagcatgacc aacggctact ggaactggat ccggaagttc 120
cccggcaagg ccctcgagta catgggctac atcacctaca gcggcagcac ctactacaac 180
cccagcctga agggccggat cagcatcacc cggaacacca gcaagaacca gtactacctg 240
accctgtcca gcgtg 255
<210> 124
<211> 255
<212> DNA
<213> Artificial Sequence
<220>
<223> HC1b
<400> 124
gagatgcagc tgcaggaaag cggccctggc ctggtcaaac ccagcgagac actgagcctg 60
acctgcagcg tgaccggcga cagcatgacc aacggctact ggaactggat ccggaagttc 120
cccggcaagg gcctcgagta catgggctac atcacctaca gcggcagcac ctactacaac 180
cccagcctga agggccggat cagcatcacc cggaacacca gcaagaacca gtactacctg 240
aagctgtcca gcgtg 255
<210> 125
<211> 255
<212> DNA
<213> Artificial Sequence
<220>
<223> HC2a
<400> 125
gagatgaccc tgaaagagtc cggccccacc ctggtcaaac ccacccagac cctgagcctg 60
acctgcagcg tgaccggcga gagcatgacc cagggctact ggaactggat ccggaagttc 120
cccggcaagg ccctcgagta catgggctac atcacctaca gcggcagcac ctactacaac 180
cccagcctga agggccggat cagcatcacc cggcagacca gcaagaacca gtactacctg 240
accctgtcca gcgtg 255
<210> 126
<211> 255
<212> DNA
<213> Artificial Sequence
<220>
<223> HC2b
<400> 126
gagatgaccc tgaaagagtc cggccccacc ctggtcaaac ccacccagac cctgagcctg 60
acctgcagcg tgaccggcga cagcatgacc cagggctact ggaactggat ccggaagttc 120
cccggcaagg ccctcgagta catgggctac atcacctaca gcggcagcac ctactacaac 180
cccagcctga agggccggat cagcatcacc cggaacacca gcaagaacca gtactacctg 240
accctgtcca gcgtg 255
<210> 127
<211> 255
<212> DNA
<213> Artificial Sequence
<220>
<223> HC3
<400> 127
cagatgaccc tgaaagagtc cggccccacc ctggtcaaac ccacccagac cctgagcctg 60
acctgcagcg tgtccggcga cagcatgacc aacggctact ggaactggat ccggcagttc 120
cccggcaagg ccctcgagta catgggctac atcacctaca gcggcagcac ctactacaac 180
cccagcctga agggccggat caccatcacc cgggacacca gcaagaacca gtactacctg 240
accctgagca gcgtg 255
<210> 128
<211> 255
<212> DNA
<213> Artificial Sequence
<220>
<223> HC4
<400> 128
cagatgaccc tgaaagagtc cggccccacc ctggtcaaac ccacccagac cctgagcctg 60
acctgcagcg tgtccggcga gagcatgacc cagggctact ggaactggat ccggcagttc 120
cccggcaagg ccctcgagta catgggctac atcacctaca gcggcagcac ctactacaac 180
cccagcctga agggccggat caccatcacc cggcagacca gcaagaacca gtactacctg 240
accctgagca gcgtg 255
<210> 129
<211> 255
<212> DNA
<213> Artificial Sequence
<220>
<223> HC5a
<400> 129
caggtgcagc tgcaggaaag cggccctggc ctggtcaaac ccagcgagac actgagcctg 60
acctgcaccg tgtccggcga cagcatgacc aacggctact ggaactggat ccggcagccc 120
cctggcaagg gcctcgagta catgggctac atcacctaca gcggcagcac ctactacaac 180
cccagcctga agtcccggat caccatcagc cggaacacca gcaagaacca gtacagcctg 240
aagctgagca gcgtg 255
<210> 130
<211> 255
<212> DNA
<213> Artificial Sequence
<220>
<223> HC5b
<400> 130
cagatgcagc tgcaggaaag cggccctggc ctggtcaaac ccagcgagac actgagcctg 60
acctgcaccg tgtccggcga cagcatgacc aacggctact ggaactggat ccggcagccc 120
cctggcaagg gcctcgagta catgggctac atcacctaca gcggcagcac ctactacaac 180
cccagcctga agtcccggat caccatcagc cgggacacca gcaagaacca gtacagcctg 240
aagctgagca gcgtg 255
<210> 131
<211> 255
<212> DNA
<213> Artificial Sequence
<220>
<223> HC5c
<400> 131
cagatgcagc tgcagcagag cggccctggc ctggtcaaac ccagccagac cctgagcctg 60
acctgcgcca tcagcggcga cagcatgacc aacggctact ggaactggat ccggcagagc 120
cccagcagag gcctcgagta catgggctac atcacctaca gcggcagcac ctactacgcc 180
gtgtccgtga agtcccggat caccatcaac cgggacacca gcaagaacca gtacagcctg 240
cagctgagca gcgtg 255
<210> 132
<211> 255
<212> DNA
<213> Artificial Sequence
<220>
<223> LC1a
<400> 132
gacatcaaga tgacccagag ccccagcagc ctgagcgcca gcgtgggcga cagagtgacc 60
atcacatgca aggccagcca ggacatcaac agctacctga gctggctgca gcagaagccc 120
ggcaagagcc ccaagaccct gatctaccgg gccaaccgca gcgtggacgg cgtgccaagc 180
agattttccg gcagcggcag cggccaggac tacagcctga ccatcagcag cctgcagccc 240
gaggacctgg gcatc 255
<210> 133
<211> 255
<212> DNA
<213> Artificial Sequence
<220>
<223> LC1b
<400> 133
gacatcaaga tgacccagag ccccagcagc gtgtccgtgt ctcctggcca gaccgtgacc 60
atcacatgca aggccagcca ggacatcaac agctacctga gctggctgca gcagaagccc 120
ggccagtccc ccaagaccct gatctaccgg gccaaccgca gcgtggacgg cgtgccaagc 180
agattttccg gcagcggcag cggccaggac tacagcctga ccatcagcag cctgcaggcc 240
atggacgagg gcatc 255
<210> 134
<211> 255
<212> DNA
<213> Artificial Sequence
<220>
<223> LC2
<400> 134
gacatcaaga tgacccagag ccccagcagc ctgagcgcca gcgtgggcga cagagtgacc 60
atcacatgca aggccagcca ggacatcaac agctacctga gctggctgca gcagaagccc 120
ggcaagagcc ccaagaccct gatctaccgg gcccagcgga gcgtggaagg cgtgccaagc 180
agattcagcg gcagcggctc cggccaggac tacagcctga ccatcagcag cctgcagccc 240
gaggacctgg gcatc 255
<210> 135
<211> 255
<212> DNA
<213> Artificial Sequence
<220>
<223> LC3
<400> 135
gacatcaaga tgacccagag ccccagcagc ctgagcgcca gcgtgggcga cagagtgacc 60
atcacatgca aggccagcca ggacatcaac agctacctga gctggctgca gcagaagccc 120
ggcaagagcc ccaagaccct gatctaccgg gccaaccgca gcgtggacgg cgtgccaagc 180
agattttccg gcagcggcag cggccaggac tacagcctga ccatcagcag cctgcagccc 240
gaggacctgg ccacc 255
<210> 136
<211> 255
<212> DNA
<213> Artificial Sequence
<220>
<223> LC4
<400> 136
gacatcaaga tgacccagag ccccagcagc ctgagcgcca gcgtgggcga cagagtgacc 60
atcacatgca aggccagcca ggacatcaac agctacctga gctggctgca gcagaagccc 120
ggcaagagcc ccaagaccct gatctaccgg gcccagcgga gcgtggaagg cgtgccaagc 180
agattcagcg gcagcggctc cggccaggac tacagcctga ccatcagcag cctgcagccc 240
gaggacctgg ccacc 255
<210> 137
<211> 255
<212> DNA
<213> Artificial Sequence
<220>
<223> LC5a
<400> 137
gacatccaga tgacccagag ccccagcagc ctgagcgcca gcgtgggcga cagagtgacc 60
atcacatgca aggccagcca ggacatcaac agctacctga gctggctgca gcagaagccc 120
ggcaaggccc ccaagctgct gatctaccgg gccaaccgca gcgtggacgg cgtgccaagc 180
agattttccg gcagcggctc cggcaccgac tacaccttca ccatcagcag cctgcagccc 240
gaggatatcg ccacc 255
<210> 138
<211> 255
<212> DNA
<213> Artificial Sequence
<220>
<223> LC5b
<400> 138
gacatccaga tgacccagag ccccagcagc ctgagcgcca gcgtgggcga cagagtgacc 60
atcacatgca aggccagcca ggacatcaac agctacctga gctggctgca gcagaagccc 120
ggcaaggccc ccaagaccct gatctaccgg gccaaccgca gcgtggacgg cgtgccaagc 180
agattttccg gcagcggcag cggccaggac tacaccttca ccatcagcag cctgcagccc 240
gaggatatcg ccacc 255
<210> 139
<211> 255
<212> DNA
<213> Artificial Sequence
<220>
<223> LC5c
<400> 139
gagatcgtga tgacccagag ccccgccacc ctgtctctga gccctggcga gagagccacc 60
ctgagctgca aggccagcca ggacatcaac agctacctga gctggctgca gcagaagccc 120
ggccaggccc ccagaaccct gatctaccgg gccaacagaa gcgtggacgg catccccgcc 180
agattcagcg gcagcggctc cggccaggac tacaccctga ccatcagcag cctggaaccc 240
gaggacttcg ccgtg 255
<210> 140
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> mA44 VH
<400> 140
Glu Met Gln Leu Gln Glu Ser Gly Pro Ser Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ser Val Thr Gly Asp Ser Met Thr Asn Gly
20 25 30
Tyr Trp Asn Trp Ile Arg Lys Phe Pro Gly Asn Lys Leu Glu Tyr Met
35 40 45
Gly Tyr Ile Thr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys
50 55 60
Gly Arg Ile Ser Ile Thr Arg Asn Thr Ser Lys Asn Gln Tyr Tyr Leu
65 70 75 80
Gln Leu Ser Ser Val Thr Thr Glu Asp Thr Ala Thr Tyr Tyr Cys Ala
85 90 95
Arg Trp His Tyr Gly Ser Pro Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Leu Thr Val Ser Ser
115
<210> 141
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> mA44 VL
<400> 141
Asp Ile Lys Met Thr Gln Ser Pro Ser Ser Met Tyr Ala Ser Leu Gly
1 5 10 15
Glu Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ser Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Asn Arg Ser Val Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Tyr
65 70 75 80
Glu Asp Met Gly Ile Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 142
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> mA44 VL
<400> 142
Asp Ile Lys Met Thr Gln Ser Pro Ser Ser Met Tyr Ala Ser Leu Gly
1 5 10 15
Glu Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Ser Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ser Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Asn Arg Ser Val Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Tyr
65 70 75 80
Glu Asp Met Gly Ile Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg Ala Asp Ala Ala
100 105 110
Pro Thr Val Ser Ile Phe
115
<210> 143
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> IGKV3-11-02_IGKJ4-0
<400> 143
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Arg Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Arg Ser Asn Trp Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 144
<211> 123
<212> PRT
<213> Artificial Sequence
<220>
<223> IGHV6-1-02_IGHD6-13-01_IGHJ4-02
<400> 144
Gln Val Gln Leu Gln Gln Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Ile Ser Gly Asp Ser Val Ser Ser Asn
20 25 30
Ser Ala Ala Trp Asn Trp Ile Arg Gln Ser Pro Ser Arg Gly Leu Glu
35 40 45
Trp Leu Gly Arg Thr Tyr Tyr Arg Ser Lys Trp Tyr Asn Asp Tyr Ala
50 55 60
Val Ser Val Lys Ser Arg Ile Thr Ile Asn Pro Asp Thr Ser Lys Asn
65 70 75 80
Gln Phe Ser Leu Gln Leu Asn Ser Val Thr Pro Glu Asp Thr Ala Val
85 90 95
Tyr Tyr Cys Ala Arg Gly Tyr Ser Ser Ser Trp Tyr Tyr Phe Asp Tyr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 145
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 145
Arg Ala Gln
1
<210> 146
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 146
Gly Glu Ser Met Thr Gln Gly Tyr
1 5
<210> 147
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 147
Gly Asp Ser Met Thr Gln Gly Tyr
1 5
<210> 148
<211> 117
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 148
Gln Val Lys Leu Gln Glu Ser Gly Pro Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Ser Phe Thr Asp Tyr
20 25 30
Asn Met Asn Trp Val Lys Gln Ser Lys Gly Lys Ser Leu Glu Trp Ile
35 40 45
Gly Ile Ile His Pro Asn Ser Gly Thr Thr Thr Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Val Asp Gln Ser Ser Ser Thr Ala Tyr
65 70 75 80
Leu Gln Leu Asn Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Lys Leu Arg Phe Phe Asp Tyr Trp Gly Gln Gly Thr Thr
100 105 110
Val Thr Val Ser Ser
115
<210> 149
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 149
Asp Ile Lys Leu Thr Gln Ser Pro Ser Ser Met Tyr Ala Ser Leu Gly
1 5 10 15
Glu Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Tyr Ser Tyr
20 25 30
Leu Ser Trp Phe Gln Gln Lys Pro Gly Lys Ser Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Asn Arg Leu Ile Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Tyr
65 70 75 80
Glu His Met Gly Ile Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Phe
85 90 95
Thr Phe Gly Ser Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 150
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 150
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Asn Leu Pro Cys
85 90 95
Ser Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 151
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<220>
<221> misc_feature
<222> (99)..(103)
<223> Xaa can be any naturally occurring amino acid
<400> 151
Gln Val Gln Leu Val Gln Ser Gly Ser Glu Leu Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Thr Asn Thr Gly Asn Pro Thr Tyr Ala Gln Gly Phe
50 55 60
Thr Gly Arg Phe Val Phe Ser Leu Asp Thr Ser Val Ser Thr Ala Tyr
65 70 75 80
Leu Gln Ile Ser Ser Leu Lys Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Xaa Xaa Xaa Xaa Xaa Tyr Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 152
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<220>
<221> misc_feature
<222> (99)..(103)
<223> Xaa can be any naturally occurring amino acid
<400> 152
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Asn Pro Ser Gly Gly Ser Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Xaa Xaa Xaa Xaa Xaa Tyr Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 153
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 153
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Tyr Ser Tyr
20 25 30
Leu Ser Trp Phe Gln Gln Lys Pro Gly Lys Ala Pro Lys Thr Leu Ile
35 40 45
Tyr Arg Ala Asn Arg Leu Ile Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Gln Asp Tyr Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Leu Gln Tyr Asp Glu Phe Pro Phe
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 154
<211> 117
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 154
Gln Val Gln Leu Val Gln Ser Gly Ser Glu Leu Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Ser Phe Thr Asp Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ile Ile His Pro Asn Ser Gly Thr Thr Thr Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Arg Ala Val Leu Ser Val Asp Gln Ser Val Ser Thr Ala Tyr
65 70 75 80
Leu Gln Ile Ser Ser Leu Lys Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Lys Leu Arg Phe Phe Asp Tyr Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser
115
<210> 155
<211> 117
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 155
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Ser Phe Thr Asp Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ile Ile His Pro Asn Ser Gly Thr Thr Thr Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Arg Ala Thr Leu Thr Val Asp Gln Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Lys Leu Arg Phe Phe Asp Tyr Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser
115
<210> 156
<211> 4
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<220>
<221> misc_feature
<222> (2)..(3)
<223> Xaa can be any naturally occurring amino acid
<400> 156
Glu Xaa Xaa Gln
1
<210> 157
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<220>
<221> misc_feature
<222> (2)..(2)
<223> Xaa can be any naturally occurring amino acid
<400> 157
Leu Xaa Arg
1
<210> 158
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<220>
<221> misc_feature
<222> (3)..(4)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (13)..(14)
<223> Xaa can be any naturally occurring amino acid
<400> 158
Thr Asp Xaa Xaa Arg Gln Phe Gln Ala Asp Phe Thr Xaa Xaa Ser Asp
1 5 10 15
Gln Glu Pro Leu
20
<210> 159
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 159
Thr Thr Gly Gly Glu Thr Arg Gln Gln Ile Gln
1 5 10
<210> 160
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 160
Arg His Leu
1
<210> 161
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<220>
<221> misc_feature
<222> (4)..(6)
<223> Xaa can be any naturally occurring amino acid
<400> 161
Thr Asp Met Xaa Xaa Xaa Phe Gln Ala Asp Phe Thr Ser Leu Ser Asn
1 5 10 15
Gln Glu Pro Leu His Val
20
<210> 162
<211> 402
<212> PRT
<213> cynomolgus
<400> 162
Met Gln Met Ser Pro Ala Leu Ala Cys Leu Val Leu Gly Leu Ala Phe
1 5 10 15
Val Phe Gly Glu Gly Ser Thr Val His His Pro Pro Ser Tyr Val Ala
20 25 30
His Leu Ala Ser Asp Phe Gly Val Arg Val Phe Gln Gln Val Ala Gln
35 40 45
Ala Ser Lys Asp Arg Asn Val Val Phe Ser Pro Tyr Gly Val Ala Ser
50 55 60
Val Leu Ala Met Leu Gln Leu Thr Thr Gly Gly Glu Thr Arg Gln Gln
65 70 75 80
Ile Gln Ala Ala Met Gly Phe Lys Ile Asp Asp Lys Gly Met Ala Pro
85 90 95
Ala Leu Arg His Leu Tyr Lys Glu Leu Leu Gly Pro Trp Asn Lys Asp
100 105 110
Glu Ile Ser Thr Thr Asp Ala Ile Phe Val Gln Arg Asp Leu Lys Leu
115 120 125
Val Gln Gly Phe Met Pro His Phe Phe Arg Leu Phe Arg Ser Thr Val
130 135 140
Lys Gln Val Asp Phe Ser Glu Ala Glu Arg Ala Arg Phe Ile Ile Asn
145 150 155 160
Asp Trp Val Lys Thr His Thr Lys Gly Met Ile Ser Asp Leu Leu Gly
165 170 175
Lys Gly Ala Val Asp Gln Leu Thr Arg Leu Val Leu Val Asn Ala Leu
180 185 190
Tyr Phe Asn Gly His Trp Lys Thr Pro Phe Pro Asp Ser Ser Thr His
195 200 205
Arg Arg Leu Phe His Lys Ser Asp Gly Ser Thr Val Ser Val Pro Met
210 215 220
Met Ala Gln Thr Asn Lys Phe Asn Tyr Thr Glu Phe Thr Thr Pro Asp
225 230 235 240
Gly His Tyr Tyr Asp Ile Leu Glu Leu Pro Tyr His Gly Asn Thr Leu
245 250 255
Ser Met Phe Ile Ala Ala Pro Tyr Glu Lys Gln Val Pro Leu Ser Ala
260 265 270
Leu Thr Asn Ile Leu Ser Ala Gln Leu Ile Ser His Trp Lys Gly Asn
275 280 285
Met Thr Arg Leu Pro Arg Leu Leu Val Leu Pro Lys Phe Ser Leu Glu
290 295 300
Thr Glu Val Asp Leu Arg Lys Pro Leu Glu Asn Leu Gly Met Thr Asp
305 310 315 320
Met Phe Arg Gln Phe Gln Ala Asp Phe Thr Ser Leu Ser Asn Gln Glu
325 330 335
Pro Leu His Val Ala Gln Ala Leu Gln Lys Val Lys Ile Glu Val Asn
340 345 350
Glu Ser Gly Thr Val Ala Ser Ser Ser Thr Ala Val Ile Val Ser Ala
355 360 365
Arg Met Ala Pro Glu Glu Ile Ile Met Asp Arg Pro Phe Leu Phe Val
370 375 380
Val Arg His Asn Pro Thr Gly Thr Val Leu Phe Met Gly Gln Val Met
385 390 395 400
Glu Pro
<210> 163
<211> 402
<212> PRT
<213> Mus musculus
<400> 163
Met Gln Met Ser Ser Ala Leu Ala Cys Leu Ile Leu Gly Leu Val Leu
1 5 10 15
Val Ser Gly Lys Gly Phe Thr Leu Pro Leu Arg Glu Ser His Thr Ala
20 25 30
His Gln Ala Thr Asp Phe Gly Val Lys Val Phe Gln Gln Val Val Gln
35 40 45
Ala Ser Lys Asp Arg Asn Val Val Phe Ser Pro Tyr Gly Val Ser Ser
50 55 60
Val Leu Ala Met Leu Gln Met Thr Thr Ala Gly Lys Thr Arg Arg Gln
65 70 75 80
Ile Gln Asp Ala Met Gly Phe Lys Val Asn Glu Lys Gly Thr Ala His
85 90 95
Ala Leu Arg Gln Leu Ser Lys Glu Leu Met Gly Pro Trp Asn Lys Asn
100 105 110
Glu Ile Ser Thr Ala Asp Ala Ile Phe Val Gln Arg Asp Leu Glu Leu
115 120 125
Val Gln Gly Phe Met Pro His Phe Phe Lys Leu Phe Gln Thr Met Val
130 135 140
Lys Gln Val Asp Phe Ser Glu Val Glu Arg Ala Arg Phe Ile Ile Asn
145 150 155 160
Asp Trp Val Glu Arg His Thr Lys Gly Met Ile Ser Asp Leu Leu Ala
165 170 175
Lys Gly Ala Val Asp Glu Leu Thr Arg Leu Val Leu Val Asn Ala Leu
180 185 190
Tyr Phe Ser Gly Gln Trp Lys Thr Pro Phe Leu Glu Ala Ser Thr His
195 200 205
Gln Arg Leu Phe His Lys Ser Asp Gly Ser Thr Val Ser Val Pro Met
210 215 220
Met Ala Gln Ser Asn Lys Phe Asn Tyr Thr Glu Phe Thr Thr Pro Asp
225 230 235 240
Gly Leu Glu Tyr Asp Val Val Glu Leu Pro Tyr Gln Gly Asp Thr Leu
245 250 255
Ser Met Phe Ile Ala Ala Pro Phe Glu Lys Asp Val His Leu Ser Ala
260 265 270
Leu Thr Asn Ile Leu Asp Ala Glu Leu Ile Arg Gln Trp Lys Gly Asn
275 280 285
Met Thr Arg Leu Pro Arg Leu Leu Ile Leu Pro Lys Phe Ser Leu Glu
290 295 300
Thr Glu Val Asp Leu Arg Gly Pro Leu Glu Lys Leu Gly Met Pro Asp
305 310 315 320
Met Phe Ser Ala Thr Leu Ala Asp Phe Thr Ser Leu Ser Asp Gln Glu
325 330 335
Gln Leu Ser Val Ala Gln Ala Leu Gln Lys Val Arg Ile Glu Val Asn
340 345 350
Glu Ser Gly Thr Val Ala Ser Ser Ser Thr Ala Phe Val Ile Ser Ala
355 360 365
Arg Met Ala Pro Thr Glu Met Val Ile Asp Arg Ser Phe Leu Phe Val
370 375 380
Val Arg His Asn Pro Thr Glu Thr Ile Leu Phe Met Gly Gln Val Met
385 390 395 400
Glu Pro
<210> 164
<211> 402
<212> PRT
<213> Rattus norvegicus
<400> 164
Met Gln Met Ser Ser Ala Leu Thr Cys Leu Thr Leu Gly Leu Val Leu
1 5 10 15
Val Phe Gly Lys Gly Phe Ala Ser Pro Leu Pro Glu Ser His Thr Ala
20 25 30
Gln Gln Ala Thr Asn Phe Gly Val Lys Val Phe Gln His Val Val Gln
35 40 45
Ala Ser Lys Asp Arg Asn Val Val Phe Ser Pro Tyr Gly Val Ser Ser
50 55 60
Val Leu Ala Met Leu Gln Leu Thr Thr Ala Gly Lys Thr Arg Gln Gln
65 70 75 80
Ile Gln Asp Ala Met Gly Phe Asn Ile Ser Glu Arg Gly Thr Ala Pro
85 90 95
Ala Leu Arg Lys Leu Ser Lys Glu Leu Met Gly Ser Trp Asn Lys Asn
100 105 110
Glu Ile Ser Thr Ala Asp Ala Ile Phe Val Gln Arg Asp Leu Glu Leu
115 120 125
Val Gln Gly Phe Met Pro His Phe Phe Lys Leu Phe Arg Thr Thr Val
130 135 140
Lys Gln Val Asp Phe Ser Glu Val Glu Arg Ala Arg Phe Ile Ile Asn
145 150 155 160
Asp Trp Val Glu Arg His Thr Lys Gly Met Ile Ser Asp Leu Leu Ala
165 170 175
Lys Gly Ala Val Asn Glu Leu Thr Arg Leu Val Leu Val Asn Ala Leu
180 185 190
Tyr Phe Asn Gly Gln Trp Lys Thr Pro Phe Leu Glu Ala Ser Thr His
195 200 205
Gln Arg Leu Phe His Lys Ser Asp Gly Ser Thr Ile Ser Val Pro Met
210 215 220
Met Ala Gln Asn Asn Lys Phe Asn Tyr Thr Glu Phe Thr Thr Pro Asp
225 230 235 240
Gly His Glu Tyr Asp Ile Leu Glu Leu Pro Tyr His Gly Glu Thr Leu
245 250 255
Ser Met Phe Ile Ala Ala Pro Phe Glu Lys Asp Val Pro Leu Ser Ala
260 265 270
Ile Thr Asn Ile Leu Asp Ala Glu Leu Ile Arg Gln Trp Lys Ser Asn
275 280 285
Met Thr Arg Leu Pro Arg Leu Leu Ile Leu Pro Lys Phe Ser Leu Glu
290 295 300
Thr Glu Val Asp Leu Arg Gly Pro Leu Glu Lys Leu Gly Met Thr Asp
305 310 315 320
Ile Phe Ser Ser Thr Gln Ala Asp Phe Thr Ser Leu Ser Asp Gln Glu
325 330 335
Gln Leu Ser Val Ala Gln Ala Leu Gln Lys Val Lys Ile Glu Val Asn
340 345 350
Glu Ser Gly Thr Val Ala Ser Ser Ser Thr Ala Ile Leu Val Ser Ala
355 360 365
Arg Met Ala Pro Thr Glu Met Val Leu Asp Arg Ser Phe Leu Phe Val
370 375 380
Val Arg His Asn Pro Thr Glu Thr Ile Leu Phe Met Gly Gln Leu Met
385 390 395 400
Glu Pro
<210> 165
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 165
Gln Asp Ile Tyr Ser Tyr
1 5
<210> 166
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 166
Leu Gln Tyr Asp Glu Phe Pro Phe Thr
1 5
<210> 167
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 167
Arg Ala Asn
1
<210> 168
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 168
Gly Tyr Ser Phe Thr Asp Tyr Asn
1 5
<210> 169
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 169
Leu Gln Tyr Asp Glu Phe Pro Phe Thr
1 5
<210> 170
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 170
Arg Ala Asn
1
Claims (15)
- (a) 중쇄 구조형성(framework) 영역 및 중쇄 가변 영역(상기 중쇄 가변 영역은 서열 번호 34를 포함하는 중쇄 CDR1 영역, 서열 번호 33을 포함하는 중쇄 CDR2 영역, 및 서열 번호 32를 포함하는 중쇄 CDR3 영역을 포함함); 및
(b) 경쇄 구조형성 영역 및 경쇄 가변 영역(상기 경쇄 가변 영역은 서열 번호 37을 포함하는 경쇄 CDR1 영역, 서열 번호 145를 포함하는 경쇄 CDR2 영역, 및 서열 번호 35를 포함하는 경쇄 CDR3 영역을 포함함)을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체. - (a) 중쇄 구조형성 영역 및 서열 번호 86을 포함하는 중쇄 가변 영역; 및
(b) 경쇄 구조형성 영역 및 서열 번호 93을 포함하는 경쇄 가변 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체. - (a) 제2항의 항체의 상기 중쇄 가변 영역과 적어도 95% 동일한 중쇄 가변 영역; 및/또는
(b) 제2항의 항체의 상기 경쇄 가변 영역과 적어도 95% 동일한 경쇄 가변 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체. - 제1항의 항체와 본질적으로 동일한 에피토프에 결합하는 단리된 단일클론 항체.
- (a) 중쇄 구조형성 영역 및 중쇄 가변 영역(상기 중쇄 가변 영역은 서열 번호 34를 포함하는 중쇄 CDR1 영역, 서열 번호 33을 포함하는 중쇄 CDR2 영역 및 서열 번호 32를 포함하는 중쇄 CDR3 영역을 포함함); 및
(b) 경쇄 구조형성 영역 및 경쇄 가변 영역(상기 경쇄 가변 영역은 서열 번호 37을 포함하는 경쇄 CDR1 영역, 서열 번호 36을 포함하는 경쇄 CDR2 영역, 및 서열 번호 35를 포함하는 경쇄 CDR3 영역을 포함함)을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체. - 제5항에 있어서, 상기 중쇄 가변 영역이 서열 번호 6을 포함하고, 상기 경쇄 가변 영역이 서열 번호 7을 포함하는 항체.
- 제5항의 항체와 본질적으로 동일한 에피토프에 결합하는 단리된 단일클론 항체.
- (a) 서열 번호 82를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편 및 서열 번호 91을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편;
(b) 서열 번호 83을 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 92를 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편;
(c) 서열 번호 84를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 93을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편;
(d) 서열 번호 85를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 91을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편;
(e) 서열 번호 85를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 93을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편;
(f) 서열 번호 86을 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 94를 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편;
(g) 서열 번호 87을 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 또는 이의 항원 결합 단편, 및 서열 번호 95를 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편;
(h) 서열 번호 88을 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 96을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편;
(i) 서열 번호 89를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 97을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편;
(j) 서열 번호 90을 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 98을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편;
(l) 서열 번호 86을 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 95를 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편;
(m) 서열 번호 89를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 93을 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편; 또는
(n) 서열 번호 89를 포함하는 중쇄 가변 영역을 가지는 중쇄, 또는 이의 항원 결합 단편, 및 서열 번호 95를 포함하는 경쇄 가변 영역을 가지는 경쇄, 또는 이의 항원 결합 단편을 포함하는, 인간 PAI-1에 특이적으로 결합하는 인간화 단일클론 항체. - (a) 서열 번호 22를 포함하는 중쇄 CDR1 영역, 서열 번호 21을 포함하는 중쇄 CDR2 영역, 및 서열 번호 20을 포함하는 중쇄 CDR3 영역을 포함하는 중쇄 가변 영역; 및 서열 번호 25를 포함하는 경쇄 CDR1 영역, 서열 번호 24를 포함하는 경쇄 CDR2 영역, 및 서열 번호 23을 포함하는 경쇄 CDR3 영역을 포함하는 경쇄 가변 영역,
(b) 서열 번호 28을 포함하는 중쇄 CDR1 영역, 서열 번호 27을 포함하는 중쇄 CDR2 영역, 및 서열 번호 26을 포함하는 중쇄 CDR3 영역을 포함하는 중쇄 가변 영역; 및 서열 번호 31을 포함하는 경쇄 CDR1 영역, 서열 번호 30을 포함하는 경쇄 CDR2 영역, 및 서열 번호 29를 포함하는 경쇄 CDR3 영역을 포함하는 경쇄 가변 영역,
(c) 서열 번호 40을 포함하는 중쇄 CDR1 영역, 서열 번호 39를 포함하는 중쇄 CDR2 영역, 및 서열 번호 38을 포함하는 중쇄 CDR3 영역을 포함하는 중쇄 가변 영역; 및 서열 번호 43을 포함하는 경쇄 CDR1 영역, 서열 번호 42를 포함하는 경쇄 CDR2 영역, 및 서열 번호 41을 포함하는 경쇄 CDR3 영역을 포함하는 경쇄 가변 영역,
(d) 서열 번호 46을 포함하는 중쇄 CDR1 영역, 서열 번호 45를 포함하는 중쇄 CDR2 영역, 및 서열 번호 44를 포함하는 중쇄 CDR3 영역을 포함하는 중쇄 가변 영역; 및 서열 번호 49를 포함하는 경쇄 CDR1 영역, 서열 번호 48을 포함하는 경쇄 CDR2 영역, 및 서열 번호 47을 포함하는 경쇄 CDR3 영역을 포함하는 경쇄 가변 영역,
(e) 서열 번호 52를 포함하는 중쇄 CDR1 영역, 서열 번호 51을 포함하는 중쇄 CDR2 영역, 및 서열 번호 50을 포함하는 중쇄 CDR3 영역을 포함하는 중쇄 가변 영역; 및 서열 번호 55를 포함하는 경쇄 CDR1 영역, 서열 번호 54를 포함하는 경쇄 CDR2 영역, 및 서열 번호 53을 포함하는 경쇄 CDR3 영역을 포함하는 경쇄 가변 영역,
(f) 서열 번호 58을 포함하는 중쇄 CDR1 영역, 서열 번호 57을 포함하는 중쇄 CDR2 영역, 및 서열 번호 56을 포함하는 중쇄 CDR3 영역을 포함하는 중쇄 가변 영역; 및 서열 번호 61을 포함하는 경쇄 CDR1 영역, 서열 번호 60을 포함하는 경쇄 CDR2 영역, 및 서열 번호 59를 포함하는 경쇄 CDR3 영역을 포함하는 경쇄 가변 영역,
(g) 서열 번호 64를 포함하는 중쇄 CDR1 영역, 서열 번호 63을 포함하는 중쇄 CDR2 영역, 및 서열 번호 62를 포함하는 중쇄 CDR3 영역을 포함하는 중쇄 가변 영역; 및 서열 번호 67을 포함하는 경쇄 CDR1 영역, 서열 번호 66을 포함하는 경쇄 CDR2 영역, 및 서열 번호 65를 포함하는 경쇄 CDR3 영역을 포함하는 경쇄 가변 영역,
(h) 서열 번호 70을 포함하는 중쇄 CDR1 영역, 서열 번호 69를 포함하는 중쇄 CDR2 영역, 및 서열 번호 68을 포함하는 중쇄 CDR3 영역을 포함하는 중쇄 가변 영역; 및 서열 번호 73을 포함하는 경쇄 CDR1 영역, 서열 번호 72를 포함하는 경쇄 CDR2 영역, 및 서열 번호 71을 포함하는 경쇄 CDR3 영역을 포함하는 경쇄 가변 영역; 또는
(i) 서열 번호 76을 포함하는 중쇄 CDR1 영역, 서열 번호 75를 포함하는 중쇄 CDR2 영역, 및 서열 번호 74를 포함하는 중쇄 CDR3 영역을 포함하는 중쇄 가변 영역; 및 서열 번호 79를 포함하는 경쇄 CDR1 영역, 서열 번호 78을 포함하는 경쇄 CDR2 영역, 및 서열 번호 77을 포함하는 경쇄 CDR3 영역을 포함하는 경쇄 가변 영역을 포함하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체. - 제8항 또는 제9항의 인간화 단일클론 항체와 본질적으로 동일한 PAI-1 상의 에피토프에 결합하는, PAI-1에 특이적으로 결합하는 단리된 단일클론 항체.
- 플리스민 생성을 회복하는 방법으로서, 필요로 하는 대상체에게 약학적 유효량의 PAI-1 항체를 경구로, 주사용 용액에 의해 비경구로, 흡입으로, 또는 국소로 투여하는 단계를 포함하는 방법.
- 제11항에 있어서, 상기 방법이 증가된 수준의 섬유증 조직을 포함하는 병태를 치료하는 방법.
- 제12항에 있어서, 상기 병태가 섬유증, 피부 섬유증, 전신성 경화증, 폐 섬유증, 특발성 폐 섬유증, 간질성 폐 질환, 만성 폐 질환, 간 섬유증, 신장 섬유증, 만성 신장 질환, 혈전증, 혈관 및 동맥 혈전증, 심부 정맥 혈전증, 말초 사지 허혈, 파종혈관내응고 혈전증, 혈전 용해를 동반하는 그리고 동반하지 않는 급성 허혈성 뇌졸중, 또는 스텐트 재협착인, 방법.
- 제11항, 제12항, 또는 제13항에 있어서, 상기 PAI-1 항체가 전술된 청구항 중 어느 한 항의 항체를 포함하는 방법.
- 환자에게 경구로, 주사용 용액에 의해 비경구로, 흡입으로, 또는 국소로 투여하는 단계를 포함하는, PAI-1의 증가된 수준 또는 PAI-1에 대한 증가된 감수성에 의해 유발되는 병태를 치료하기 위한 약제의 제조를 위한 약학적 유효량의 PAI-1 항체의 용도.
Applications Claiming Priority (5)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201361865451P | 2013-08-13 | 2013-08-13 | |
| US61/865,451 | 2013-08-13 | ||
| EP14305757 | 2014-05-22 | ||
| EP14305757.8 | 2014-05-22 | ||
| PCT/US2014/050896 WO2015023752A1 (en) | 2013-08-13 | 2014-08-13 | Antibodies to plasminogen activator inhibitor-1 (pai-1) and uses thereof |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20160035077A true KR20160035077A (ko) | 2016-03-30 |
Family
ID=56194112
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020167005652A KR20160035077A (ko) | 2013-08-13 | 2014-08-13 | 플라스미노겐 활성인자 저해제-1(pai-1)에 대한 항체 및 그의 용도 |
Country Status (17)
| Country | Link |
|---|---|
| US (4) | US9845363B2 (ko) |
| JP (4) | JP6696899B2 (ko) |
| KR (1) | KR20160035077A (ko) |
| CN (2) | CN112142845A (ko) |
| BR (1) | BR112016002753A2 (ko) |
| CL (1) | CL2016000324A1 (ko) |
| CR (2) | CR20190319A (ko) |
| DO (1) | DOP2016000042A (ko) |
| EA (1) | EA033403B1 (ko) |
| ES (1) | ES2770507T3 (ko) |
| HK (1) | HK1221725A1 (ko) |
| MX (2) | MX2016001851A (ko) |
| PE (1) | PE20160244A1 (ko) |
| PH (1) | PH12016500239A1 (ko) |
| SG (1) | SG10201710013RA (ko) |
| TN (1) | TN2016000048A1 (ko) |
| UA (1) | UA118267C2 (ko) |
Families Citing this family (21)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CA3008186A1 (en) | 2015-12-18 | 2017-06-22 | Talengen International Limited | Novel method for preventing and treating angiocardiopathy |
| CA3008686C (en) | 2015-12-18 | 2023-03-14 | Talengen International Limited | Method for preventing and treating diabetic nephropathy |
| DK3395353T3 (da) | 2015-12-18 | 2024-03-11 | Talengen Int Ltd | Plasminogen til anvendelse til behandling eller forebyggelse af diabetes-mellitus-nerveskade |
| WO2017101870A1 (zh) | 2015-12-18 | 2017-06-22 | 深圳瑞健生命科学研究院有限公司 | 一种预防或治疗糖尿病性视网膜病变的方法 |
| WO2018107694A1 (zh) * | 2016-12-15 | 2018-06-21 | 深圳瑞健生命科学研究院有限公司 | 一种预防和治疗皮肤纤维化的方法 |
| TWI763680B (zh) * | 2016-12-15 | 2022-05-11 | 大陸商深圳瑞健生命科學硏究院有限公司 | 纖溶酶原在製備藥物中的用途 |
| WO2018107686A1 (zh) | 2016-12-15 | 2018-06-21 | 深圳瑞健生命科学研究院有限公司 | 一种治疗动脉粥样硬化及其并发症的方法 |
| US20190343930A1 (en) * | 2016-12-15 | 2019-11-14 | Talengen International Limited | Method for preventing and treating pathological renal tissue injury |
| CN108210900A (zh) * | 2016-12-15 | 2018-06-29 | 深圳瑞健生命科学研究院有限公司 | 预防和治疗肥胖症的药物及其用途 |
| EP3556379A4 (en) | 2016-12-15 | 2020-08-19 | Talengen International Limited | METHOD AND DRUG FOR THE PREVENTION AND TREATMENT OF OBESITY |
| CN108210897A (zh) * | 2016-12-15 | 2018-06-29 | 深圳瑞健生命科学研究院有限公司 | 预防和治疗肺纤维化的药物及其用途 |
| CA3067890A1 (en) | 2017-06-19 | 2018-12-27 | Talengen International Limited | Method for regulating and controling glp-1/glp-1r and drug |
| TW201904990A (zh) * | 2017-06-23 | 2019-02-01 | 美商波麥堤克生物治療股份有限公司 | 與pai-1過表現相關之病狀的纖維蛋白溶酶原治療 |
| CN110662765B (zh) | 2017-08-03 | 2023-09-29 | 艾利妥 | 抗cd33抗体及其使用方法 |
| WO2021066062A1 (ja) | 2019-09-30 | 2021-04-08 | 株式会社レナサイエンス | 免疫チェックポイント分子の発現抑制剤 |
| US11827709B2 (en) * | 2019-12-05 | 2023-11-28 | Seagen Inc. | Anti-AVB6 antibodies and antibody-drug conjugates |
| EP4094776A4 (en) * | 2020-02-11 | 2023-05-17 | Talengen International Limited | METHODS AND MEDICATIONS FOR THE TREATMENT OF VIRAL PENMONIA |
| CN111072780B (zh) * | 2020-03-24 | 2020-06-23 | 深圳汉盛汇融再生医学科技有限公司 | 白血病干细胞抑制剂及其在治疗慢性粒细胞白血病中的应用 |
| CN114605550B (zh) * | 2020-12-08 | 2023-09-22 | 东莞市朋志生物科技有限公司 | 抗ca19-9的抗体、其应用和检测ca19-9的试剂盒 |
| CN114686441B (zh) * | 2020-12-31 | 2023-11-03 | 广州万孚生物技术股份有限公司 | 一种能分泌tPAI-C单克隆抗体的杂交瘤细胞株及其应用 |
| CN114686442B (zh) * | 2020-12-31 | 2023-11-07 | 广州万孚生物技术股份有限公司 | 一种杂交瘤细胞株及其应用 |
Family Cites Families (74)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US4741900A (en) | 1982-11-16 | 1988-05-03 | Cytogen Corporation | Antibody-metal ion complexes |
| GB8308235D0 (en) | 1983-03-25 | 1983-05-05 | Celltech Ltd | Polypeptides |
| US4816567A (en) | 1983-04-08 | 1989-03-28 | Genentech, Inc. | Recombinant immunoglobin preparations |
| US5807715A (en) | 1984-08-27 | 1998-09-15 | The Board Of Trustees Of The Leland Stanford Junior University | Methods and transformed mammalian lymphocyte cells for producing functional antigen-binding protein including chimeric immunoglobulin |
| DK319685A (da) * | 1985-07-12 | 1987-01-13 | Fonden Til Fremme Af Eksperime | Monoklonale antistoffer, fremgangsmaade til frembringelse af antistofferne, hybridomaceller, der producerer antistofferne, og anvendelse af antistofferne |
| GB8607679D0 (en) | 1986-03-27 | 1986-04-30 | Winter G P | Recombinant dna product |
| US5225539A (en) | 1986-03-27 | 1993-07-06 | Medical Research Council | Recombinant altered antibodies and methods of making altered antibodies |
| DE3883899T3 (de) | 1987-03-18 | 1999-04-22 | Sb2 Inc | Geänderte antikörper. |
| JP2750732B2 (ja) * | 1988-04-04 | 1998-05-13 | 帝人株式会社 | ヒトプラスミノーゲンアクティベーターインヒビター・1−ヒト組織プラスミノーゲンアクティベーター複合体の免疫学的測定方法 |
| DE68921982T4 (de) | 1988-06-14 | 1996-04-25 | Cetus Oncology Corp | Kupplungsmittel und sterisch gehinderte, mit disulfid gebundene konjugate daraus. |
| US6780613B1 (en) | 1988-10-28 | 2004-08-24 | Genentech, Inc. | Growth hormone variants |
| US5530101A (en) | 1988-12-28 | 1996-06-25 | Protein Design Labs, Inc. | Humanized immunoglobulins |
| ZA902949B (en) | 1989-05-05 | 1992-02-26 | Res Dev Foundation | A novel antibody delivery system for biological response modifiers |
| US5460785A (en) | 1989-08-09 | 1995-10-24 | Rhomed Incorporated | Direct labeling of antibodies and other protein with metal ions |
| GB8928874D0 (en) | 1989-12-21 | 1990-02-28 | Celltech Ltd | Humanised antibodies |
| US5314995A (en) | 1990-01-22 | 1994-05-24 | Oncogen | Therapeutic interleukin-2-antibody based fusion proteins |
| JP3319594B2 (ja) | 1990-03-20 | 2002-09-03 | ザ・トラスティーズ・オブ・コランビア・ユニバーシティー・イン・ザ・シティー・オブ・ニューヨーク | 定常領域の代わりに受容体結合性リガンドを有するキメラ抗体 |
| ATE218889T1 (de) | 1990-11-09 | 2002-06-15 | Stephen D Gillies | Cytokine immunokonjugate |
| EP0519596B1 (en) | 1991-05-17 | 2005-02-23 | Merck & Co. Inc. | A method for reducing the immunogenicity of antibody variable domains |
| ES2136092T3 (es) | 1991-09-23 | 1999-11-16 | Medical Res Council | Procedimientos para la produccion de anticuerpos humanizados. |
| US5639641A (en) | 1992-09-09 | 1997-06-17 | Immunogen Inc. | Resurfacing of rodent antibodies |
| DK85193D0 (da) * | 1993-07-16 | 1993-07-16 | Cancerforskningsfondet Af 1989 | Suppression of inhibitors |
| GB9422383D0 (en) | 1994-11-05 | 1995-01-04 | Wellcome Found | Antibodies |
| US5739277A (en) | 1995-04-14 | 1998-04-14 | Genentech Inc. | Altered polypeptides with increased half-life |
| US5869046A (en) | 1995-04-14 | 1999-02-09 | Genentech, Inc. | Altered polypeptides with increased half-life |
| US6096871A (en) | 1995-04-14 | 2000-08-01 | Genentech, Inc. | Polypeptides altered to contain an epitope from the Fc region of an IgG molecule for increased half-life |
| US6121022A (en) | 1995-04-14 | 2000-09-19 | Genentech, Inc. | Altered polypeptides with increased half-life |
| GB9524973D0 (en) | 1995-12-06 | 1996-02-07 | Lynxvale Ltd | Viral vectors |
| JPH1014592A (ja) * | 1996-07-04 | 1998-01-20 | Tanabe Seiyaku Co Ltd | ラットpai−1に対するモノクローナル抗体 |
| CA2262405A1 (en) | 1996-08-02 | 1998-02-12 | Bristol-Myers Squibb Company | A method for inhibiting immunoglobulin-induced toxicity resulting from the use of immunoglobulins in therapy and in vivo diagnosis |
| WO1998023289A1 (en) | 1996-11-27 | 1998-06-04 | The General Hospital Corporation | MODULATION OF IgG BINDING TO FcRn |
| US6277375B1 (en) | 1997-03-03 | 2001-08-21 | Board Of Regents, The University Of Texas System | Immunoglobulin-like domains with increased half-lives |
| AU736549B2 (en) | 1997-05-21 | 2001-08-02 | Merck Patent Gesellschaft Mit Beschrankter Haftung | Method for the production of non-immunogenic proteins |
| JP2002510481A (ja) | 1998-04-02 | 2002-04-09 | ジェネンテック・インコーポレーテッド | 抗体変異体及びその断片 |
| US6242195B1 (en) | 1998-04-02 | 2001-06-05 | Genentech, Inc. | Methods for determining binding of an analyte to a receptor |
| US6194551B1 (en) | 1998-04-02 | 2001-02-27 | Genentech, Inc. | Polypeptide variants |
| US6528624B1 (en) | 1998-04-02 | 2003-03-04 | Genentech, Inc. | Polypeptide variants |
| GB9809951D0 (en) | 1998-05-08 | 1998-07-08 | Univ Cambridge Tech | Binding molecules |
| WO2000009560A2 (en) | 1998-08-17 | 2000-02-24 | Abgenix, Inc. | Generation of modified molecules with increased serum half-lives |
| EP1006183A1 (en) | 1998-12-03 | 2000-06-07 | Max-Planck-Gesellschaft zur Förderung der Wissenschaften e.V. | Recombinant soluble Fc receptors |
| CN1202128C (zh) | 1998-12-08 | 2005-05-18 | 拜奥威神有限公司 | 修饰蛋白的免疫原性 |
| US6737056B1 (en) | 1999-01-15 | 2004-05-18 | Genentech, Inc. | Polypeptide variants with altered effector function |
| PL209786B1 (pl) | 1999-01-15 | 2011-10-31 | Genentech Inc | Przeciwciało zawierające wariant regionu Fc ludzkiej IgG1, przeciwciało wiążące czynnik wzrostu śródbłonka naczyń oraz immunoadhezyna |
| MY133346A (en) | 1999-03-01 | 2007-11-30 | Biogen Inc | Kit for radiolabeling ligands with yttrium-90 |
| US20020102208A1 (en) | 1999-03-01 | 2002-08-01 | Paul Chinn | Radiolabeling kit and binding assay |
| WO2002034776A2 (en) | 2000-10-26 | 2002-05-02 | K.U.Leuven Research And Development | Epitopes of pai-1 |
| GB0029407D0 (en) | 2000-12-01 | 2001-01-17 | Affitech As | Product |
| ES2649037T3 (es) | 2000-12-12 | 2018-01-09 | Medimmune, Llc | Moléculas con semividas prolongadas, composiciones y usos de las mismas |
| US20040002587A1 (en) | 2002-02-20 | 2004-01-01 | Watkins Jeffry D. | Fc region variants |
| US20040132101A1 (en) | 2002-09-27 | 2004-07-08 | Xencor | Optimized Fc variants and methods for their generation |
| US20040018557A1 (en) | 2002-03-01 | 2004-01-29 | Immunomedics, Inc. | Bispecific antibody point mutations for enhancing rate of clearance |
| AU2003301809A1 (en) * | 2002-05-13 | 2004-06-07 | Children's Hospital Los Angeles | Treatment and prevention of abnormal scar formation in keloids and other cutaneous or internal wounds or lesions |
| US7538195B2 (en) * | 2002-06-14 | 2009-05-26 | Immunogen Inc. | Anti-IGF-I receptor antibody |
| ATE536188T1 (de) | 2002-08-14 | 2011-12-15 | Macrogenics Inc | Fcgammariib-spezifische antikörper und verfahren zur verwendung davon |
| ATE541857T1 (de) | 2002-09-27 | 2012-02-15 | Xencor Inc | Optimierte fc-varianten und herstellungsverfahren dafür |
| JP4768439B2 (ja) | 2002-10-15 | 2011-09-07 | アボット バイオセラピューティクス コーポレイション | 変異誘発による抗体のFcRn結合親和力又は血清半減期の改変 |
| JP2006524039A (ja) | 2003-01-09 | 2006-10-26 | マクロジェニクス,インコーポレーテッド | 変異型Fc領域を含む抗体の同定および作製ならびにその利用法 |
| AU2004266159A1 (en) | 2003-08-22 | 2005-03-03 | Biogen Idec Ma Inc. | Improved antibodies having altered effector function and methods for making the same |
| GB0324368D0 (en) | 2003-10-17 | 2003-11-19 | Univ Cambridge Tech | Polypeptides including modified constant regions |
| WO2005047327A2 (en) | 2003-11-12 | 2005-05-26 | Biogen Idec Ma Inc. | NEONATAL Fc RECEPTOR (FcRn)-BINDING POLYPEPTIDE VARIANTS, DIMERIC Fc BINDING PROTEINS AND METHODS RELATED THERETO |
| US20050249723A1 (en) | 2003-12-22 | 2005-11-10 | Xencor, Inc. | Fc polypeptides with novel Fc ligand binding sites |
| BRPI0506771A (pt) | 2004-01-12 | 2007-05-22 | Applied Molecular Evolution | anticorpo, e, composição farmacêutica |
| AU2005227326B2 (en) | 2004-03-24 | 2009-12-03 | Xencor, Inc. | Immunoglobulin variants outside the Fc region |
| WO2005123780A2 (en) | 2004-04-09 | 2005-12-29 | Protein Design Labs, Inc. | Alteration of fcrn binding affinities or serum half-lives of antibodies by mutagenesis |
| WO2006085967A2 (en) | 2004-07-09 | 2006-08-17 | Xencor, Inc. | OPTIMIZED ANTI-CD20 MONOCONAL ANTIBODIES HAVING Fc VARIANTS |
| EP3342782B1 (en) | 2004-07-15 | 2022-08-17 | Xencor, Inc. | Optimized fc variants |
| WO2006047350A2 (en) | 2004-10-21 | 2006-05-04 | Xencor, Inc. | IgG IMMUNOGLOBULIN VARIANTS WITH OPTIMIZED EFFECTOR FUNCTION |
| US20090202524A1 (en) | 2007-10-31 | 2009-08-13 | Alcon Research, Ltd. | Pai-1 expression and activity inhibitors for the treatment of ocular disorders |
| PT2195023T (pt) | 2007-08-29 | 2018-06-08 | Sanofi Sa | Anticorpos anti-cxcr5 humanizados, seus derivados e suas utilizações |
| US7771720B2 (en) | 2007-09-07 | 2010-08-10 | Cisthera, Inc. | Humanized PAI-1 antibodies |
| US7981415B2 (en) | 2007-09-07 | 2011-07-19 | Cisthera, Inc. | Humanized PAI-1 antibodies |
| US8410251B2 (en) * | 2008-06-20 | 2013-04-02 | National University Corporation Okayama University | Antibody against calcified globule and use of the same |
| US20100254979A1 (en) * | 2009-03-06 | 2010-10-07 | Cisthera, Incorporated | Humanized PAI-1 Antibodies and Uses Thereof |
| EP2566890A4 (en) * | 2010-05-03 | 2013-11-20 | Abbvie Inc | ANTI-PAI-1 ANTIBODIES AND METHOD FOR THEIR USE |
-
2014
- 2014-08-13 KR KR1020167005652A patent/KR20160035077A/ko not_active Application Discontinuation
- 2014-08-13 UA UAA201602278A patent/UA118267C2/uk unknown
- 2014-08-13 ES ES14761451T patent/ES2770507T3/es active Active
- 2014-08-13 JP JP2016534815A patent/JP6696899B2/ja active Active
- 2014-08-13 EA EA201690377A patent/EA033403B1/ru not_active IP Right Cessation
- 2014-08-13 MX MX2016001851A patent/MX2016001851A/es unknown
- 2014-08-13 BR BR112016002753A patent/BR112016002753A2/pt not_active Application Discontinuation
- 2014-08-13 CN CN202010898460.6A patent/CN112142845A/zh active Pending
- 2014-08-13 PE PE2016000243A patent/PE20160244A1/es unknown
- 2014-08-13 TN TN2016000048A patent/TN2016000048A1/en unknown
- 2014-08-13 CN CN201480055157.2A patent/CN105705520B/zh active Active
- 2014-08-13 CR CR20190319A patent/CR20190319A/es unknown
- 2014-08-13 US US14/911,429 patent/US9845363B2/en active Active
- 2014-08-13 SG SG10201710013RA patent/SG10201710013RA/en unknown
-
2016
- 2016-02-03 PH PH12016500239A patent/PH12016500239A1/en unknown
- 2016-02-03 DO DO2016000042A patent/DOP2016000042A/es unknown
- 2016-02-10 CL CL2016000324A patent/CL2016000324A1/es unknown
- 2016-02-10 MX MX2019008803A patent/MX2019008803A/es unknown
- 2016-03-07 CR CR20160117A patent/CR20160117A/es unknown
- 2016-08-17 HK HK16109838.9A patent/HK1221725A1/zh unknown
-
2017
- 2017-11-10 US US15/809,935 patent/US20180155444A1/en not_active Abandoned
-
2019
- 2019-08-06 US US16/533,048 patent/US20190359729A1/en not_active Abandoned
-
2020
- 2020-04-23 JP JP2020076578A patent/JP6907380B2/ja active Active
-
2021
- 2021-06-29 JP JP2021107196A patent/JP7223069B2/ja active Active
-
2022
- 2022-06-17 US US17/843,777 patent/US20230015181A1/en active Pending
-
2023
- 2023-02-02 JP JP2023014335A patent/JP2023055852A/ja active Pending
Also Published As
| Publication number | Publication date |
|---|---|
| MX2019008803A (es) | 2019-09-16 |
| US20230015181A1 (en) | 2023-01-19 |
| US20190359729A1 (en) | 2019-11-28 |
| HK1221725A1 (zh) | 2017-06-09 |
| CR20160117A (es) | 2016-05-20 |
| JP2016529255A (ja) | 2016-09-23 |
| PE20160244A1 (es) | 2016-05-10 |
| CL2016000324A1 (es) | 2016-10-21 |
| MX2016001851A (es) | 2016-05-16 |
| CN112142845A (zh) | 2020-12-29 |
| CN105705520A (zh) | 2016-06-22 |
| PH12016500239A1 (en) | 2016-05-16 |
| BR112016002753A2 (pt) | 2017-11-21 |
| ES2770507T3 (es) | 2020-07-01 |
| JP6907380B2 (ja) | 2021-07-21 |
| US9845363B2 (en) | 2017-12-19 |
| CR20190319A (es) | 2019-08-29 |
| US20180155444A1 (en) | 2018-06-07 |
| TN2016000048A1 (en) | 2017-07-05 |
| CN105705520B (zh) | 2020-09-25 |
| DOP2016000042A (es) | 2016-04-04 |
| JP2020125333A (ja) | 2020-08-20 |
| SG10201710013RA (en) | 2018-01-30 |
| JP7223069B2 (ja) | 2023-02-15 |
| JP2023055852A (ja) | 2023-04-18 |
| JP6696899B2 (ja) | 2020-05-20 |
| EA033403B1 (ru) | 2019-10-31 |
| JP2021155440A (ja) | 2021-10-07 |
| US20160200831A1 (en) | 2016-07-14 |
| EA201690377A1 (ru) | 2016-06-30 |
| UA118267C2 (uk) | 2018-12-26 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP6907380B2 (ja) | プラスミノーゲン活性化因子阻害剤−1(pai−1)に対する抗体及びその使用 | |
| EP3033359B1 (en) | Antibodies to plasminogen activator inhibitor-1 (pai-1) and uses thereof | |
| JP7411609B2 (ja) | ブラジキニンb1受容体リガンドに対する抗体 | |
| CA2745317C (en) | Antibodies against tissue factor pathway inhibitor | |
| US20160297892A1 (en) | Novel Methods and Antibodies for Treating Coagulapathy | |
| CN111683965A (zh) | 用于治疗骨折或骨缺损的gremlin-1抑制剂 | |
| JP2010533732A (ja) | プロテアーゼ活性化受容体−1(par1)のアンタゴニスト抗体 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| WITB | Written withdrawal of application |