JP6420147B2 - メバロン酸、イソプレノイド前駆体、及びイソプレンの産生におけるホスホケトラーゼの使用 - Google Patents
メバロン酸、イソプレノイド前駆体、及びイソプレンの産生におけるホスホケトラーゼの使用 Download PDFInfo
- Publication number
- JP6420147B2 JP6420147B2 JP2014534810A JP2014534810A JP6420147B2 JP 6420147 B2 JP6420147 B2 JP 6420147B2 JP 2014534810 A JP2014534810 A JP 2014534810A JP 2014534810 A JP2014534810 A JP 2014534810A JP 6420147 B2 JP6420147 B2 JP 6420147B2
- Authority
- JP
- Japan
- Prior art keywords
- polypeptide
- phosphoketolase
- nucleic acid
- heterologous nucleic
- isoprene
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
- 108010004621 phosphoketolase Proteins 0.000 title claims description 519
- KJTLQQUUPVSXIM-ZCFIWIBFSA-N (R)-mevalonic acid Chemical compound OCC[C@](O)(C)CC(O)=O KJTLQQUUPVSXIM-ZCFIWIBFSA-N 0.000 title claims description 323
- KJTLQQUUPVSXIM-UHFFFAOYSA-N DL-mevalonic acid Natural products OCCC(O)(C)CC(O)=O KJTLQQUUPVSXIM-UHFFFAOYSA-N 0.000 title claims description 323
- RRHGJUQNOFWUDK-UHFFFAOYSA-N Isoprene Chemical compound CC(=C)C=C RRHGJUQNOFWUDK-UHFFFAOYSA-N 0.000 title claims description 220
- 150000003505 terpenes Chemical class 0.000 title claims description 180
- 238000004519 manufacturing process Methods 0.000 title claims description 97
- 239000002243 precursor Substances 0.000 title claims description 64
- 210000004027 cell Anatomy 0.000 claims description 508
- 150000007523 nucleic acids Chemical class 0.000 claims description 438
- 102000039446 nucleic acids Human genes 0.000 claims description 411
- 108020004707 nucleic acids Proteins 0.000 claims description 411
- 108090000765 processed proteins & peptides Proteins 0.000 claims description 410
- 102000004196 processed proteins & peptides Human genes 0.000 claims description 386
- 229920001184 polypeptide Polymers 0.000 claims description 379
- 230000000694 effects Effects 0.000 claims description 152
- 230000037361 pathway Effects 0.000 claims description 128
- ZSLZBFCDCINBPY-ZSJPKINUSA-N acetyl-CoA Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCSC(=O)C)O[C@H]1N1C2=NC=NC(N)=C2N=C1 ZSLZBFCDCINBPY-ZSJPKINUSA-N 0.000 claims description 115
- 241001608472 Bifidobacterium longum Species 0.000 claims description 95
- 229940009291 bifidobacterium longum Drugs 0.000 claims description 77
- LIPOUNRJVLNBCD-UHFFFAOYSA-N acetyl dihydrogen phosphate Chemical compound CC(=O)OP(O)(O)=O LIPOUNRJVLNBCD-UHFFFAOYSA-N 0.000 claims description 66
- 102000004190 Enzymes Human genes 0.000 claims description 65
- 108090000790 Enzymes Proteins 0.000 claims description 65
- 241000186604 Lactobacillus reuteri Species 0.000 claims description 62
- 238000000034 method Methods 0.000 claims description 61
- 229940001882 lactobacillus reuteri Drugs 0.000 claims description 57
- 241000187392 Streptomyces griseus Species 0.000 claims description 56
- 230000014509 gene expression Effects 0.000 claims description 55
- OJFDKHTZOUZBOS-CITAKDKDSA-N acetoacetyl-CoA Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCSC(=O)CC(=O)C)O[C@H]1N1C2=NC=NC(N)=C2N=C1 OJFDKHTZOUZBOS-CITAKDKDSA-N 0.000 claims description 50
- 241000193401 Clostridium acetobutylicum Species 0.000 claims description 47
- FNZLKVNUWIIPSJ-RFZPGFLSSA-N D-xylulose 5-phosphate Chemical compound OCC(=O)[C@@H](O)[C@H](O)COP(O)(O)=O FNZLKVNUWIIPSJ-RFZPGFLSSA-N 0.000 claims description 36
- CBIDRCWHNCKSTO-UHFFFAOYSA-N prenyl diphosphate Chemical compound CC(C)=CCO[P@](O)(=O)OP(O)(O)=O CBIDRCWHNCKSTO-UHFFFAOYSA-N 0.000 claims description 36
- 108010075483 isoprene synthase Proteins 0.000 claims description 35
- GSXOAOHZAIYLCY-UHFFFAOYSA-N D-F6P Natural products OCC(=O)C(O)C(O)C(O)COP(O)(O)=O GSXOAOHZAIYLCY-UHFFFAOYSA-N 0.000 claims description 34
- BGWGXPAPYGQALX-ARQDHWQXSA-N beta-D-fructofuranose 6-phosphate Chemical compound OC[C@@]1(O)O[C@H](COP(O)(O)=O)[C@@H](O)[C@@H]1O BGWGXPAPYGQALX-ARQDHWQXSA-N 0.000 claims description 33
- 239000013612 plasmid Substances 0.000 claims description 33
- 238000012258 culturing Methods 0.000 claims description 30
- NUHSROFQTUXZQQ-UHFFFAOYSA-N isopentenyl diphosphate Chemical compound CC(=C)CCO[P@](O)(=O)OP(O)(O)=O NUHSROFQTUXZQQ-UHFFFAOYSA-N 0.000 claims description 28
- NGHMDNPXVRFFGS-IUYQGCFVSA-N D-erythrose 4-phosphate Chemical compound O=C[C@H](O)[C@H](O)COP(O)(O)=O NGHMDNPXVRFFGS-IUYQGCFVSA-N 0.000 claims description 25
- LXJXRIRHZLFYRP-VKHMYHEASA-L (R)-2-Hydroxy-3-(phosphonooxy)-propanal Natural products O=C[C@H](O)COP([O-])([O-])=O LXJXRIRHZLFYRP-VKHMYHEASA-L 0.000 claims description 24
- LXJXRIRHZLFYRP-VKHMYHEASA-N D-glyceraldehyde 3-phosphate Chemical compound O=C[C@H](O)COP(O)(O)=O LXJXRIRHZLFYRP-VKHMYHEASA-N 0.000 claims description 24
- CABVTRNMFUVUDM-VRHQGPGLSA-N (3S)-3-hydroxy-3-methylglutaryl-CoA Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCSC(=O)C[C@@](O)(CC(O)=O)C)O[C@H]1N1C2=NC=NC(N)=C2N=C1 CABVTRNMFUVUDM-VRHQGPGLSA-N 0.000 claims description 23
- 241000588724 Escherichia coli Species 0.000 claims description 22
- 230000001965 increasing effect Effects 0.000 claims description 21
- 240000006024 Lactobacillus plantarum Species 0.000 claims description 20
- 230000001580 bacterial effect Effects 0.000 claims description 20
- 241001134770 Bifidobacterium animalis Species 0.000 claims description 19
- 235000013965 Lactobacillus plantarum Nutrition 0.000 claims description 19
- 229940072205 lactobacillus plantarum Drugs 0.000 claims description 19
- SIGQQUBJQXSAMW-ZCFIWIBFSA-N (R)-5-diphosphomevalonic acid Chemical compound OC(=O)C[C@@](O)(C)CCOP(O)(=O)OP(O)(O)=O SIGQQUBJQXSAMW-ZCFIWIBFSA-N 0.000 claims description 18
- 229910019142 PO4 Inorganic materials 0.000 claims description 17
- 229940118852 bifidobacterium animalis Drugs 0.000 claims description 17
- 239000010452 phosphate Substances 0.000 claims description 17
- 241000194033 Enterococcus Species 0.000 claims description 16
- 241000186012 Bifidobacterium breve Species 0.000 claims description 15
- 235000011180 diphosphates Nutrition 0.000 claims description 15
- 210000000349 chromosome Anatomy 0.000 claims description 13
- JSNRRGGBADWTMC-UHFFFAOYSA-N alpha-farnesene Natural products CC(C)=CCCC(C)=CCCC(=C)C=C JSNRRGGBADWTMC-UHFFFAOYSA-N 0.000 claims description 12
- 108010092060 Acetate kinase Proteins 0.000 claims description 11
- 241000894006 Bacteria Species 0.000 claims description 11
- 241000193403 Clostridium Species 0.000 claims description 11
- 230000001939 inductive effect Effects 0.000 claims description 11
- 229920001550 polyprenyl Polymers 0.000 claims description 11
- 125000001185 polyprenyl group Polymers 0.000 claims description 11
- 241000186000 Bifidobacterium Species 0.000 claims description 10
- 108700023175 Phosphate acetyltransferases Proteins 0.000 claims description 10
- 210000005253 yeast cell Anatomy 0.000 claims description 10
- OKZYCXHTTZZYSK-ZCFIWIBFSA-N (R)-5-phosphomevalonic acid Chemical compound OC(=O)C[C@@](O)(C)CCOP(O)(O)=O OKZYCXHTTZZYSK-ZCFIWIBFSA-N 0.000 claims description 9
- 241000196324 Embryophyta Species 0.000 claims description 9
- AJPADPZSRRUGHI-RFZPGFLSSA-N 1-deoxy-D-xylulose 5-phosphate Chemical compound CC(=O)[C@@H](O)[C@H](O)COP(O)(O)=O AJPADPZSRRUGHI-RFZPGFLSSA-N 0.000 claims description 8
- GLZPCOQZEFWAFX-UHFFFAOYSA-N Geraniol Chemical compound CC(C)=CCCC(C)=CCO GLZPCOQZEFWAFX-UHFFFAOYSA-N 0.000 claims description 8
- 241000168036 Populus alba Species 0.000 claims description 8
- UAHWPYUMFXYFJY-UHFFFAOYSA-N beta-myrcene Chemical compound CC(C)=CCCC(=C)C=C UAHWPYUMFXYFJY-UHFFFAOYSA-N 0.000 claims description 8
- XMGQYMWWDOXHJM-UHFFFAOYSA-N limonene Chemical compound CC(=C)C1CCC(C)=CC1 XMGQYMWWDOXHJM-UHFFFAOYSA-N 0.000 claims description 8
- CDOSHBSSFJOMGT-UHFFFAOYSA-N linalool Chemical compound CC(C)=CCCC(C)(O)C=C CDOSHBSSFJOMGT-UHFFFAOYSA-N 0.000 claims description 8
- NDVASEGYNIMXJL-UHFFFAOYSA-N sabinene Chemical compound C=C1CCC2(C(C)C)C1C2 NDVASEGYNIMXJL-UHFFFAOYSA-N 0.000 claims description 8
- CXENHBSYCFFKJS-UHFFFAOYSA-N α-farnesene Chemical compound CC(C)=CCCC(C)=CCC=C(C)C=C CXENHBSYCFFKJS-UHFFFAOYSA-N 0.000 claims description 8
- 241000235070 Saccharomyces Species 0.000 claims description 7
- 241000219000 Populus Species 0.000 claims description 6
- 235000010575 Pueraria lobata Nutrition 0.000 claims description 6
- 241000124033 Salix Species 0.000 claims description 6
- 235000014469 Bacillus subtilis Nutrition 0.000 claims description 5
- 241000186660 Lactobacillus Species 0.000 claims description 5
- 241000187432 Streptomyces coelicolor Species 0.000 claims description 5
- 241000187398 Streptomyces lividans Species 0.000 claims description 5
- 241000520244 Tatumella citrea Species 0.000 claims description 5
- 229940039696 lactobacillus Drugs 0.000 claims description 5
- 229930003658 monoterpene Natural products 0.000 claims description 5
- 230000002829 reductive effect Effects 0.000 claims description 5
- 150000003648 triterpenes Chemical class 0.000 claims description 5
- FQTLCLSUCSAZDY-UHFFFAOYSA-N (+) E(S) nerolidol Natural products CC(C)=CCCC(C)=CCCC(C)(O)C=C FQTLCLSUCSAZDY-UHFFFAOYSA-N 0.000 claims description 4
- NDVASEGYNIMXJL-NXEZZACHSA-N (+)-sabinene Natural products C=C1CC[C@@]2(C(C)C)[C@@H]1C2 NDVASEGYNIMXJL-NXEZZACHSA-N 0.000 claims description 4
- QEBNYNLSCGVZOH-NFAWXSAZSA-N (+)-valencene Chemical compound C1C[C@@H](C(C)=C)C[C@@]2(C)[C@H](C)CCC=C21 QEBNYNLSCGVZOH-NFAWXSAZSA-N 0.000 claims description 4
- WTARULDDTDQWMU-RKDXNWHRSA-N (+)-β-pinene Chemical compound C1[C@H]2C(C)(C)[C@@H]1CCC2=C WTARULDDTDQWMU-RKDXNWHRSA-N 0.000 claims description 4
- BBPXZLJCPUPNGH-CMKODMSKSA-N (-)-Abietadiene Chemical compound CC1(C)CCC[C@]2(C)[C@@H](CCC(C(C)C)=C3)C3=CC[C@H]21 BBPXZLJCPUPNGH-CMKODMSKSA-N 0.000 claims description 4
- WTARULDDTDQWMU-IUCAKERBSA-N (-)-Nopinene Natural products C1[C@@H]2C(C)(C)[C@H]1CCC2=C WTARULDDTDQWMU-IUCAKERBSA-N 0.000 claims description 4
- CRDAMVZIKSXKFV-FBXUGWQNSA-N (2-cis,6-cis)-farnesol Chemical compound CC(C)=CCC\C(C)=C/CC\C(C)=C/CO CRDAMVZIKSXKFV-FBXUGWQNSA-N 0.000 claims description 4
- 239000000260 (2E,6E)-3,7,11-trimethyldodeca-2,6,10-trien-1-ol Substances 0.000 claims description 4
- 239000001890 (2R)-8,8,8a-trimethyl-2-prop-1-en-2-yl-1,2,3,4,6,7-hexahydronaphthalene Substances 0.000 claims description 4
- 239000001490 (3R)-3,7-dimethylocta-1,6-dien-3-ol Substances 0.000 claims description 4
- JSNRRGGBADWTMC-QINSGFPZSA-N (E)-beta-Farnesene Natural products CC(C)=CCC\C(C)=C/CCC(=C)C=C JSNRRGGBADWTMC-QINSGFPZSA-N 0.000 claims description 4
- OJISWRZIEWCUBN-QIRCYJPOSA-N (E,E,E)-geranylgeraniol Chemical compound CC(C)=CCC\C(C)=C\CC\C(C)=C\CC\C(C)=C\CO OJISWRZIEWCUBN-QIRCYJPOSA-N 0.000 claims description 4
- CDOSHBSSFJOMGT-JTQLQIEISA-N (R)-linalool Natural products CC(C)=CCC[C@@](C)(O)C=C CDOSHBSSFJOMGT-JTQLQIEISA-N 0.000 claims description 4
- JXBSHSBNOVLGHF-UHFFFAOYSA-N 10-cis-Dihydrofarnesen Natural products CC=C(C)CCC=C(C)CCC=C(C)C JXBSHSBNOVLGHF-UHFFFAOYSA-N 0.000 claims description 4
- BBPXZLJCPUPNGH-UHFFFAOYSA-N Abietadien Natural products CC1(C)CCCC2(C)C(CCC(C(C)C)=C3)C3=CCC21 BBPXZLJCPUPNGH-UHFFFAOYSA-N 0.000 claims description 4
- 244000063299 Bacillus subtilis Species 0.000 claims description 4
- 239000005792 Geraniol Substances 0.000 claims description 4
- GLZPCOQZEFWAFX-YFHOEESVSA-N Geraniol Natural products CC(C)=CCC\C(C)=C/CO GLZPCOQZEFWAFX-YFHOEESVSA-N 0.000 claims description 4
- FQTLCLSUCSAZDY-ATGUSINASA-N Nerolidol Chemical compound CC(C)=CCC\C(C)=C\CC[C@](C)(O)C=C FQTLCLSUCSAZDY-ATGUSINASA-N 0.000 claims description 4
- WTARULDDTDQWMU-UHFFFAOYSA-N Pseudopinene Natural products C1C2C(C)(C)C1CCC2=C WTARULDDTDQWMU-UHFFFAOYSA-N 0.000 claims description 4
- 229930014549 abietadiene Natural products 0.000 claims description 4
- XCPQUQHBVVXMRQ-UHFFFAOYSA-N alpha-Fenchene Natural products C1CC2C(=C)CC1C2(C)C XCPQUQHBVVXMRQ-UHFFFAOYSA-N 0.000 claims description 4
- VYBREYKSZAROCT-UHFFFAOYSA-N alpha-myrcene Natural products CC(=C)CCCC(=C)C=C VYBREYKSZAROCT-UHFFFAOYSA-N 0.000 claims description 4
- KQAZVFVOEIRWHN-UHFFFAOYSA-N alpha-thujene Natural products CC1=CCC2(C(C)C)C1C2 KQAZVFVOEIRWHN-UHFFFAOYSA-N 0.000 claims description 4
- HMTAHNDPLDKYJT-CBBWQLFWSA-N amorpha-4,11-diene Chemical compound C1=C(C)CC[C@H]2[C@H](C)CC[C@@H](C(C)=C)[C@H]21 HMTAHNDPLDKYJT-CBBWQLFWSA-N 0.000 claims description 4
- HMTAHNDPLDKYJT-UHFFFAOYSA-N amorphadiene Natural products C1=C(C)CCC2C(C)CCC(C(C)=C)C21 HMTAHNDPLDKYJT-UHFFFAOYSA-N 0.000 claims description 4
- YSNRTFFURISHOU-UHFFFAOYSA-N beta-farnesene Natural products C=CC(C)CCC=C(C)CCC=C(C)C YSNRTFFURISHOU-UHFFFAOYSA-N 0.000 claims description 4
- 229930006722 beta-pinene Natural products 0.000 claims description 4
- 229930004069 diterpene Natural products 0.000 claims description 4
- 125000000567 diterpene group Chemical group 0.000 claims description 4
- 229930002886 farnesol Natural products 0.000 claims description 4
- 229940043259 farnesol Drugs 0.000 claims description 4
- LCWMKIHBLJLORW-UHFFFAOYSA-N gamma-carene Natural products C1CC(=C)CC2C(C)(C)C21 LCWMKIHBLJLORW-UHFFFAOYSA-N 0.000 claims description 4
- 229940113087 geraniol Drugs 0.000 claims description 4
- XWRJRXQNOHXIOX-UHFFFAOYSA-N geranylgeraniol Natural products CC(C)=CCCC(C)=CCOCC=C(C)CCC=C(C)C XWRJRXQNOHXIOX-UHFFFAOYSA-N 0.000 claims description 4
- OJISWRZIEWCUBN-UHFFFAOYSA-N geranylnerol Natural products CC(C)=CCCC(C)=CCCC(C)=CCCC(C)=CCO OJISWRZIEWCUBN-UHFFFAOYSA-N 0.000 claims description 4
- 235000001510 limonene Nutrition 0.000 claims description 4
- 229940087305 limonene Drugs 0.000 claims description 4
- 229930007744 linalool Natural products 0.000 claims description 4
- 150000002773 monoterpene derivatives Chemical class 0.000 claims description 4
- 235000002577 monoterpenes Nutrition 0.000 claims description 4
- WASNIKZYIWZQIP-AWEZNQCLSA-N nerolidol Natural products CC(=CCCC(=CCC[C@@H](O)C=C)C)C WASNIKZYIWZQIP-AWEZNQCLSA-N 0.000 claims description 4
- 150000003097 polyterpenes Chemical class 0.000 claims description 4
- 229930006696 sabinene Natural products 0.000 claims description 4
- RXHIKAIVEMAPRU-JRIGQVHBSA-N sequiterpene Natural products C1=C(C)[C@@H](OC(C)=O)[C@H](O)[C@@]2(O)[C@H](C)CC[C@@H](C(C)=C)[C@H]21 RXHIKAIVEMAPRU-JRIGQVHBSA-N 0.000 claims description 4
- 229930004725 sesquiterpene Natural products 0.000 claims description 4
- 150000004354 sesquiterpene derivatives Chemical class 0.000 claims description 4
- 150000003535 tetraterpenes Chemical class 0.000 claims description 4
- 235000009657 tetraterpenes Nutrition 0.000 claims description 4
- CRDAMVZIKSXKFV-UHFFFAOYSA-N trans-Farnesol Natural products CC(C)=CCCC(C)=CCCC(C)=CCO CRDAMVZIKSXKFV-UHFFFAOYSA-N 0.000 claims description 4
- WCTNXGFHEZQHDR-UHFFFAOYSA-N valencene Natural products C1CC(C)(C)C2(C)CC(C(=C)C)CCC2=C1 WCTNXGFHEZQHDR-UHFFFAOYSA-N 0.000 claims description 4
- 241000218982 Populus nigra Species 0.000 claims description 3
- 241001600128 Populus tremula x Populus alba Species 0.000 claims description 3
- 240000004923 Populus tremuloides Species 0.000 claims description 3
- 235000011263 Populus tremuloides Nutrition 0.000 claims description 3
- 241000218976 Populus trichocarpa Species 0.000 claims description 3
- 241000219780 Pueraria Species 0.000 claims description 3
- 241000913776 Pueraria montana Species 0.000 claims description 3
- 241000235015 Yarrowia lipolytica Species 0.000 claims description 3
- XPPKVPWEQAFLFU-UHFFFAOYSA-J diphosphate(4-) Chemical compound [O-]P([O-])(=O)OP([O-])([O-])=O XPPKVPWEQAFLFU-UHFFFAOYSA-J 0.000 claims description 3
- 101001074421 Bacillus subtilis (strain 168) Polyketide biosynthesis 3-hydroxy-3-methylglutaryl-ACP synthase PksG Proteins 0.000 claims description 2
- 241000899950 Salix glauca Species 0.000 claims description 2
- 241000186146 Brevibacterium Species 0.000 claims 1
- 241000219781 Pueraria montana var. lobata Species 0.000 claims 1
- 229930006737 car-3-ene Natural products 0.000 claims 1
- 229930007796 carene Natural products 0.000 claims 1
- BQOFWKZOCNGFEC-UHFFFAOYSA-N carene Chemical compound C1C(C)=CCC2C(C)(C)C12 BQOFWKZOCNGFEC-UHFFFAOYSA-N 0.000 claims 1
- 125000001151 peptidyl group Chemical group 0.000 claims 1
- 229930006978 terpinene Natural products 0.000 claims 1
- 150000003507 terpinene derivatives Chemical class 0.000 claims 1
- 108090000623 proteins and genes Proteins 0.000 description 109
- 241000194030 Enterococcus gallinarum Species 0.000 description 62
- 229940088598 enzyme Drugs 0.000 description 59
- 101100507308 Enterococcus faecalis mvaS gene Proteins 0.000 description 44
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 44
- 238000006243 chemical reaction Methods 0.000 description 44
- 241000203619 Nocardiopsis dassonvillei Species 0.000 description 42
- 125000003275 alpha amino acid group Chemical group 0.000 description 39
- 102000004169 proteins and genes Human genes 0.000 description 38
- QTBSBXVTEAMEQO-UHFFFAOYSA-N Acetic acid Chemical compound CC(O)=O QTBSBXVTEAMEQO-UHFFFAOYSA-N 0.000 description 36
- 241000178317 Ferrimonas balearica Species 0.000 description 33
- 239000008103 glucose Substances 0.000 description 32
- BPHPUYQFMNQIOC-NXRLNHOXSA-N isopropyl beta-D-thiogalactopyranoside Chemical compound CC(C)S[C@@H]1O[C@H](CO)[C@H](O)[C@H](O)[C@H]1O BPHPUYQFMNQIOC-NXRLNHOXSA-N 0.000 description 30
- 239000002773 nucleotide Substances 0.000 description 30
- 125000003729 nucleotide group Chemical group 0.000 description 30
- 108091028043 Nucleic acid sequence Proteins 0.000 description 29
- 238000000855 fermentation Methods 0.000 description 29
- 230000004151 fermentation Effects 0.000 description 29
- 230000002194 synthesizing effect Effects 0.000 description 27
- 235000001014 amino acid Nutrition 0.000 description 26
- 235000018102 proteins Nutrition 0.000 description 26
- 229910052799 carbon Inorganic materials 0.000 description 24
- 239000002609 medium Substances 0.000 description 24
- OKTJSMMVPCPJKN-UHFFFAOYSA-N Carbon Chemical compound [C] OKTJSMMVPCPJKN-UHFFFAOYSA-N 0.000 description 23
- 241000424623 Nostoc punctiforme Species 0.000 description 23
- 238000011144 upstream manufacturing Methods 0.000 description 22
- 108030002854 Acetoacetyl-CoA synthases Proteins 0.000 description 21
- 239000000047 product Substances 0.000 description 21
- 241000866650 Lactobacillus paraplantarum Species 0.000 description 20
- LTYOQGRJFJAKNA-KKIMTKSISA-N Malonyl CoA Natural products S(C(=O)CC(=O)O)CCNC(=O)CCNC(=O)[C@@H](O)C(CO[P@](=O)(O[P@](=O)(OC[C@H]1[C@@H](OP(=O)(O)O)[C@@H](O)[C@@H](n2c3ncnc(N)c3nc2)O1)O)O)(C)C LTYOQGRJFJAKNA-KKIMTKSISA-N 0.000 description 20
- LTYOQGRJFJAKNA-DVVLENMVSA-N malonyl-CoA Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCSC(=O)CC(O)=O)O[C@H]1N1C2=NC=NC(N)=C2N=C1 LTYOQGRJFJAKNA-DVVLENMVSA-N 0.000 description 20
- 230000015572 biosynthetic process Effects 0.000 description 19
- 239000000758 substrate Substances 0.000 description 17
- 241001340632 Mucilaginibacter paludis Species 0.000 description 16
- 241001468227 Streptomyces avermitilis Species 0.000 description 16
- 241000203780 Thermobifida fusca Species 0.000 description 16
- 241000194032 Enterococcus faecalis Species 0.000 description 15
- 241000207201 Gardnerella vaginalis Species 0.000 description 15
- 108700040132 Mevalonate kinases Proteins 0.000 description 15
- 241000520272 Pantoea Species 0.000 description 15
- 241001478271 Rahnella aquatilis Species 0.000 description 15
- 241000190950 Rhodopseudomonas palustris Species 0.000 description 15
- 150000001413 amino acids Chemical class 0.000 description 15
- -1 isoprenoid compounds Chemical class 0.000 description 15
- 102000002678 mevalonate kinase Human genes 0.000 description 15
- 238000006467 substitution reaction Methods 0.000 description 15
- 241000194031 Enterococcus faecium Species 0.000 description 14
- 239000000203 mixture Substances 0.000 description 14
- 241001522957 Enterococcus casseliflavus Species 0.000 description 13
- 108090000895 Hydroxymethylglutaryl CoA Reductases Proteins 0.000 description 13
- 102000004286 Hydroxymethylglutaryl CoA Reductases Human genes 0.000 description 13
- 229940024606 amino acid Drugs 0.000 description 13
- 101150031187 fba gene Proteins 0.000 description 13
- 101150108901 fbaA gene Proteins 0.000 description 13
- 108010000775 Hydroxymethylglutaryl-CoA synthase Proteins 0.000 description 12
- 102100028888 Hydroxymethylglutaryl-CoA synthase, cytoplasmic Human genes 0.000 description 12
- 238000004458 analytical method Methods 0.000 description 12
- XPPKVPWEQAFLFU-UHFFFAOYSA-N diphosphoric acid Chemical compound OP(O)(=O)OP(O)(O)=O XPPKVPWEQAFLFU-UHFFFAOYSA-N 0.000 description 12
- 230000002708 enhancing effect Effects 0.000 description 12
- 241000894007 species Species 0.000 description 11
- 238000003786 synthesis reaction Methods 0.000 description 11
- OKZYCXHTTZZYSK-UHFFFAOYSA-N 3-hydroxy-3-methyl-5-phosphonooxypentanoic acid Chemical compound OC(=O)CC(O)(C)CCOP(O)(O)=O OKZYCXHTTZZYSK-UHFFFAOYSA-N 0.000 description 10
- 241000187747 Streptomyces Species 0.000 description 10
- 229940032049 enterococcus faecalis Drugs 0.000 description 10
- 241000178316 Ferrimonas Species 0.000 description 9
- 108010068561 Fructose-Bisphosphate Aldolase Proteins 0.000 description 9
- 102000001390 Fructose-Bisphosphate Aldolase Human genes 0.000 description 9
- 241000186806 Listeria grayi Species 0.000 description 9
- 102000002932 Thiolase Human genes 0.000 description 9
- 108060008225 Thiolase Proteins 0.000 description 9
- 125000002777 acetyl group Chemical group [H]C([H])([H])C(*)=O 0.000 description 9
- 230000002538 fungal effect Effects 0.000 description 9
- 238000004128 high performance liquid chromatography Methods 0.000 description 9
- 238000000338 in vitro Methods 0.000 description 9
- 108010030844 2-methylcitrate synthase Proteins 0.000 description 8
- 241001415963 Balearica Species 0.000 description 8
- 108020004530 Transaldolase Proteins 0.000 description 8
- 102000014701 Transketolase Human genes 0.000 description 8
- 108010043652 Transketolase Proteins 0.000 description 8
- 125000000539 amino acid group Chemical group 0.000 description 8
- 230000006696 biosynthetic metabolic pathway Effects 0.000 description 8
- 101150063369 mvaS gene Proteins 0.000 description 8
- 102100031126 6-phosphogluconolactonase Human genes 0.000 description 7
- 108010029731 6-phosphogluconolactonase Proteins 0.000 description 7
- 108010071536 Citrate (Si)-synthase Proteins 0.000 description 7
- 102000006732 Citrate synthase Human genes 0.000 description 7
- 101100190555 Dictyostelium discoideum pkgB gene Proteins 0.000 description 7
- 101150039774 GAPA1 gene Proteins 0.000 description 7
- 108010018962 Glucosephosphate Dehydrogenase Proteins 0.000 description 7
- 101100433987 Latilactobacillus sakei subsp. sakei (strain 23K) ackA1 gene Proteins 0.000 description 7
- 101100066407 Mus musculus Fbxo3 gene Proteins 0.000 description 7
- 101100282114 Pseudomonas aeruginosa (strain UCBPP-PA14) gap2 gene Proteins 0.000 description 7
- 101100453320 Pyrococcus furiosus (strain ATCC 43587 / DSM 3638 / JCM 8422 / Vc1) pfkC gene Proteins 0.000 description 7
- 108060007030 Ribulose-phosphate 3-epimerase Proteins 0.000 description 7
- 241000187180 Streptomyces sp. Species 0.000 description 7
- 101100029403 Synechocystis sp. (strain PCC 6803 / Kazusa) pfkA2 gene Proteins 0.000 description 7
- 101150024271 TKT gene Proteins 0.000 description 7
- 101150006213 ackA gene Proteins 0.000 description 7
- 230000001419 dependent effect Effects 0.000 description 7
- 230000006870 function Effects 0.000 description 7
- 230000004927 fusion Effects 0.000 description 7
- 101150073818 gap gene Proteins 0.000 description 7
- 101150091570 gapA gene Proteins 0.000 description 7
- 238000010353 genetic engineering Methods 0.000 description 7
- 101150106096 gltA gene Proteins 0.000 description 7
- 101150042350 gltA2 gene Proteins 0.000 description 7
- 102000006602 glyceraldehyde-3-phosphate dehydrogenase Human genes 0.000 description 7
- 108020004445 glyceraldehyde-3-phosphate dehydrogenase Proteins 0.000 description 7
- 230000003834 intracellular effect Effects 0.000 description 7
- 230000035772 mutation Effects 0.000 description 7
- 101150038284 pfkA gene Proteins 0.000 description 7
- 101150004013 pfkA1 gene Proteins 0.000 description 7
- 101150060387 pfp gene Proteins 0.000 description 7
- 238000003752 polymerase chain reaction Methods 0.000 description 7
- 102000004688 ribulosephosphate 3-epimerase Human genes 0.000 description 7
- 101150003316 rpiA gene Proteins 0.000 description 7
- 101150106193 tal gene Proteins 0.000 description 7
- 101150014795 tktA gene Proteins 0.000 description 7
- 101150078419 zwf gene Proteins 0.000 description 7
- 101100508888 Bacillus subtilis (strain 168) iolJ gene Proteins 0.000 description 6
- 101100280057 Brucella abortus (strain 2308) eryI gene Proteins 0.000 description 6
- 101100138542 Cupriavidus necator (strain ATCC 17699 / DSM 428 / KCTC 22496 / NCIMB 10442 / H16 / Stanier 337) phbH gene Proteins 0.000 description 6
- 101100299477 Cupriavidus necator (strain ATCC 17699 / DSM 428 / KCTC 22496 / NCIMB 10442 / H16 / Stanier 337) phbI gene Proteins 0.000 description 6
- KTVPXOYAKDPRHY-SOOFDHNKSA-N D-ribofuranose 5-phosphate Chemical compound OC1O[C@H](COP(O)(O)=O)[C@@H](O)[C@H]1O KTVPXOYAKDPRHY-SOOFDHNKSA-N 0.000 description 6
- 101100010747 Escherichia coli (strain K12) epd gene Proteins 0.000 description 6
- 101150098454 GAPA2 gene Proteins 0.000 description 6
- 101150036652 GAPB gene Proteins 0.000 description 6
- 101100335749 Halobacterium salinarum (strain ATCC 700922 / JCM 11081 / NRC-1) gap gene Proteins 0.000 description 6
- 101710161955 Mannitol-specific phosphotransferase enzyme IIA component Proteins 0.000 description 6
- 101100010672 Mycobacterium leprae (strain TN) dxs gene Proteins 0.000 description 6
- 241000194017 Streptococcus Species 0.000 description 6
- 239000002253 acid Substances 0.000 description 6
- 238000007792 addition Methods 0.000 description 6
- 101150001544 crr gene Proteins 0.000 description 6
- GNGACRATGGDKBX-UHFFFAOYSA-N dihydroxyacetone phosphate Chemical compound OCC(=O)COP(O)(O)=O GNGACRATGGDKBX-UHFFFAOYSA-N 0.000 description 6
- 101150105921 eutD gene Proteins 0.000 description 6
- 101150050376 fbaB gene Proteins 0.000 description 6
- 101150111583 fda gene Proteins 0.000 description 6
- 239000000499 gel Substances 0.000 description 6
- 230000037353 metabolic pathway Effects 0.000 description 6
- 101150100557 pfkB gene Proteins 0.000 description 6
- 108010071189 phosphoenolpyruvate-glucose phosphotransferase Proteins 0.000 description 6
- 101150045242 ptsH gene Proteins 0.000 description 6
- 101150118630 ptsI gene Proteins 0.000 description 6
- 101150060189 rpiB gene Proteins 0.000 description 6
- 101150071019 tktB gene Proteins 0.000 description 6
- YKFLAYDHMOASIY-UHFFFAOYSA-N γ-terpinene Chemical compound CC(C)C1=CCC(C)=CC1 YKFLAYDHMOASIY-UHFFFAOYSA-N 0.000 description 6
- 102000004195 Isomerases Human genes 0.000 description 5
- 108090000769 Isomerases Proteins 0.000 description 5
- 102000007079 Peptide Fragments Human genes 0.000 description 5
- 108010033276 Peptide Fragments Proteins 0.000 description 5
- 244000046146 Pueraria lobata Species 0.000 description 5
- LCTONWCANYUPML-UHFFFAOYSA-M Pyruvate Chemical compound CC(=O)C([O-])=O LCTONWCANYUPML-UHFFFAOYSA-M 0.000 description 5
- 102000007382 Ribose-5-phosphate isomerase Human genes 0.000 description 5
- 238000009825 accumulation Methods 0.000 description 5
- 230000009471 action Effects 0.000 description 5
- 239000003242 anti bacterial agent Substances 0.000 description 5
- 239000012620 biological material Substances 0.000 description 5
- 230000002255 enzymatic effect Effects 0.000 description 5
- 239000012634 fragment Substances 0.000 description 5
- 230000006872 improvement Effects 0.000 description 5
- 244000005700 microbiome Species 0.000 description 5
- NBIIXXVUZAFLBC-UHFFFAOYSA-K phosphate Chemical compound [O-]P([O-])([O-])=O NBIIXXVUZAFLBC-UHFFFAOYSA-K 0.000 description 5
- 108020005610 ribose 5-phosphate isomerase Proteins 0.000 description 5
- 239000000523 sample Substances 0.000 description 5
- 238000002415 sodium dodecyl sulfate polyacrylamide gel electrophoresis Methods 0.000 description 5
- 239000012130 whole-cell lysate Substances 0.000 description 5
- 241000186015 Bifidobacterium longum subsp. infantis Species 0.000 description 4
- 101710088194 Dehydrogenase Proteins 0.000 description 4
- 241000884274 Enterococcus gallinarum EG2 Species 0.000 description 4
- 241001069991 Listeria grayi DSM 20601 Species 0.000 description 4
- 241000205274 Methanosarcina mazei Species 0.000 description 4
- 102100024279 Phosphomevalonate kinase Human genes 0.000 description 4
- FNZLKVNUWIIPSJ-UHFFFAOYSA-N Rbl5P Natural products OCC(=O)C(O)C(O)COP(O)(O)=O FNZLKVNUWIIPSJ-UHFFFAOYSA-N 0.000 description 4
- QAOWNCQODCNURD-UHFFFAOYSA-N Sulfuric acid Chemical compound OS(O)(=O)=O QAOWNCQODCNURD-UHFFFAOYSA-N 0.000 description 4
- 230000001186 cumulative effect Effects 0.000 description 4
- 230000000670 limiting effect Effects 0.000 description 4
- 230000004060 metabolic process Effects 0.000 description 4
- 239000002207 metabolite Substances 0.000 description 4
- 108091000116 phosphomevalonate kinase Proteins 0.000 description 4
- 229910052698 phosphorus Inorganic materials 0.000 description 4
- 239000011574 phosphorus Substances 0.000 description 4
- 230000001105 regulatory effect Effects 0.000 description 4
- 239000000126 substance Substances 0.000 description 4
- 239000006228 supernatant Substances 0.000 description 4
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 description 3
- 108010006229 Acetyl-CoA C-acetyltransferase Proteins 0.000 description 3
- 102000005345 Acetyl-CoA C-acetyltransferase Human genes 0.000 description 3
- 241000186361 Actinobacteria <class> Species 0.000 description 3
- 241000228245 Aspergillus niger Species 0.000 description 3
- 240000006439 Aspergillus oryzae Species 0.000 description 3
- 235000002247 Aspergillus oryzae Nutrition 0.000 description 3
- FNZLKVNUWIIPSJ-UHNVWZDZSA-N D-ribulose 5-phosphate Chemical compound OCC(=O)[C@H](O)[C@H](O)COP(O)(O)=O FNZLKVNUWIIPSJ-UHNVWZDZSA-N 0.000 description 3
- ZAQJHHRNXZUBTE-WUJLRWPWSA-N D-xylulose Chemical compound OC[C@@H](O)[C@H](O)C(=O)CO ZAQJHHRNXZUBTE-WUJLRWPWSA-N 0.000 description 3
- 108020004414 DNA Proteins 0.000 description 3
- 102100038390 Diphosphomevalonate decarboxylase Human genes 0.000 description 3
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 3
- 241000282326 Felis catus Species 0.000 description 3
- 241001465754 Metazoa Species 0.000 description 3
- 241000983368 Pantoea sp. Species 0.000 description 3
- 206010035664 Pneumonia Diseases 0.000 description 3
- 241000589516 Pseudomonas Species 0.000 description 3
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 3
- 235000014680 Saccharomyces cerevisiae Nutrition 0.000 description 3
- 241001647802 Thermobifida Species 0.000 description 3
- 241000499912 Trichoderma reesei Species 0.000 description 3
- 239000007983 Tris buffer Substances 0.000 description 3
- XJLXINKUBYWONI-DQQFMEOOSA-N [[(2r,3r,4r,5r)-5-(6-aminopurin-9-yl)-3-hydroxy-4-phosphonooxyoxolan-2-yl]methoxy-hydroxyphosphoryl] [(2s,3r,4s,5s)-5-(3-carbamoylpyridin-1-ium-1-yl)-3,4-dihydroxyoxolan-2-yl]methyl phosphate Chemical compound NC(=O)C1=CC=C[N+]([C@@H]2[C@H]([C@@H](O)[C@H](COP([O-])(=O)OP(O)(=O)OC[C@@H]3[C@H]([C@@H](OP(O)(O)=O)[C@@H](O3)N3C4=NC=NC(N)=C4N=C3)O)O2)O)=C1 XJLXINKUBYWONI-DQQFMEOOSA-N 0.000 description 3
- 229940041514 candida albicans extract Drugs 0.000 description 3
- 230000008859 change Effects 0.000 description 3
- 150000001875 compounds Chemical class 0.000 description 3
- 210000004748 cultured cell Anatomy 0.000 description 3
- 238000006911 enzymatic reaction Methods 0.000 description 3
- 230000012010 growth Effects 0.000 description 3
- 239000000543 intermediate Substances 0.000 description 3
- 238000002703 mutagenesis Methods 0.000 description 3
- 231100000350 mutagenesis Toxicity 0.000 description 3
- 150000007823 ocimene derivatives Chemical class 0.000 description 3
- 108091033319 polynucleotide Proteins 0.000 description 3
- 239000002157 polynucleotide Substances 0.000 description 3
- 102000040430 polynucleotide Human genes 0.000 description 3
- 230000009467 reduction Effects 0.000 description 3
- JDTUMPKOJBQPKX-GBNDHIKLSA-N sedoheptulose 7-phosphate Chemical compound OCC(=O)[C@@H](O)[C@H](O)[C@H](O)[C@H](O)COP(O)(O)=O JDTUMPKOJBQPKX-GBNDHIKLSA-N 0.000 description 3
- 238000010561 standard procedure Methods 0.000 description 3
- 229920003051 synthetic elastomer Polymers 0.000 description 3
- XJPBRODHZKDRCB-UHFFFAOYSA-N trans-alpha-ocimene Natural products CC(=C)CCC=C(C)C=C XJPBRODHZKDRCB-UHFFFAOYSA-N 0.000 description 3
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 3
- 239000012138 yeast extract Substances 0.000 description 3
- KJTLQQUUPVSXIM-LURJTMIESA-N (S)-mevalonic acid Chemical compound OCC[C@@](O)(C)CC(O)=O KJTLQQUUPVSXIM-LURJTMIESA-N 0.000 description 2
- 101710184086 2-C-methyl-D-erythritol 2,4-cyclodiphosphate synthase Proteins 0.000 description 2
- 101710201168 2-C-methyl-D-erythritol 4-phosphate cytidylyltransferase Proteins 0.000 description 2
- 101710166309 4-diphosphocytidyl-2-C-methyl-D-erythritol kinase Proteins 0.000 description 2
- 101710139854 4-hydroxy-3-methylbut-2-en-1-yl diphosphate synthase (ferredoxin) Proteins 0.000 description 2
- 101710088071 4-hydroxy-3-methylbut-2-en-1-yl diphosphate synthase (ferredoxin), chloroplastic Proteins 0.000 description 2
- 101710086072 4-hydroxy-3-methylbut-2-en-1-yl diphosphate synthase (flavodoxin) Proteins 0.000 description 2
- 102000057234 Acyl transferases Human genes 0.000 description 2
- 108700016155 Acyl transferases Proteins 0.000 description 2
- IJGRMHOSHXDMSA-UHFFFAOYSA-N Atomic nitrogen Chemical compound N#N IJGRMHOSHXDMSA-UHFFFAOYSA-N 0.000 description 2
- 241000193830 Bacillus <bacterium> Species 0.000 description 2
- 241000194108 Bacillus licheniformis Species 0.000 description 2
- CURLTUGMZLYLDI-UHFFFAOYSA-N Carbon dioxide Chemical compound O=C=O CURLTUGMZLYLDI-UHFFFAOYSA-N 0.000 description 2
- 241000195493 Cryptophyta Species 0.000 description 2
- 241000192700 Cyanobacteria Species 0.000 description 2
- 102100037458 Dephospho-CoA kinase Human genes 0.000 description 2
- 101710183613 Diphosphomevalonate decarboxylase Proteins 0.000 description 2
- 101001078008 Escherichia coli (strain K12) 4-hydroxy-3-methylbut-2-enyl diphosphate reductase Proteins 0.000 description 2
- 241000192125 Firmicutes Species 0.000 description 2
- 229930091371 Fructose Natural products 0.000 description 2
- 239000005715 Fructose Substances 0.000 description 2
- RFSUNEUAIZKAJO-ARQDHWQXSA-N Fructose Chemical compound OC[C@H]1O[C@](O)(CO)[C@@H](O)[C@@H]1O RFSUNEUAIZKAJO-ARQDHWQXSA-N 0.000 description 2
- 241000233866 Fungi Species 0.000 description 2
- 241000207202 Gardnerella Species 0.000 description 2
- 239000004366 Glucose oxidase Substances 0.000 description 2
- 108010015776 Glucose oxidase Proteins 0.000 description 2
- PEDCQBHIVMGVHV-UHFFFAOYSA-N Glycerine Chemical compound OCC(O)CO PEDCQBHIVMGVHV-UHFFFAOYSA-N 0.000 description 2
- DHMQDGOQFOQNFH-UHFFFAOYSA-N Glycine Chemical compound NCC(O)=O DHMQDGOQFOQNFH-UHFFFAOYSA-N 0.000 description 2
- HNDVDQJCIGZPNO-YFKPBYRVSA-N L-histidine Chemical compound OC(=O)[C@@H](N)CC1=CN=CN1 HNDVDQJCIGZPNO-YFKPBYRVSA-N 0.000 description 2
- AGPKZVBTJJNPAG-WHFBIAKZSA-N L-isoleucine Chemical compound CC[C@H](C)[C@H](N)C(O)=O AGPKZVBTJJNPAG-WHFBIAKZSA-N 0.000 description 2
- COLNVLDHVKWLRT-QMMMGPOBSA-N L-phenylalanine Chemical compound OC(=O)[C@@H](N)CC1=CC=CC=C1 COLNVLDHVKWLRT-QMMMGPOBSA-N 0.000 description 2
- AYFVYJQAPQTCCC-GBXIJSLDSA-N L-threonine Chemical compound C[C@@H](O)[C@H](N)C(O)=O AYFVYJQAPQTCCC-GBXIJSLDSA-N 0.000 description 2
- QIVBCDIJIAJPQS-VIFPVBQESA-N L-tryptophane Chemical compound C1=CC=C2C(C[C@H](N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-VIFPVBQESA-N 0.000 description 2
- OUYCCCASQSFEME-QMMMGPOBSA-N L-tyrosine Chemical compound OC(=O)[C@@H](N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-QMMMGPOBSA-N 0.000 description 2
- KZSNJWFQEVHDMF-BYPYZUCNSA-N L-valine Chemical compound CC(C)[C@H](N)C(O)=O KZSNJWFQEVHDMF-BYPYZUCNSA-N 0.000 description 2
- 240000001046 Lactobacillus acidophilus Species 0.000 description 2
- 239000006137 Luria-Bertani broth Substances 0.000 description 2
- 241000894763 Nostoc punctiforme PCC 73102 Species 0.000 description 2
- 102000004316 Oxidoreductases Human genes 0.000 description 2
- 108090000854 Oxidoreductases Proteins 0.000 description 2
- NBIIXXVUZAFLBC-UHFFFAOYSA-N Phosphoric acid Chemical compound OP(O)(O)=O NBIIXXVUZAFLBC-UHFFFAOYSA-N 0.000 description 2
- OAICVXFJPJFONN-UHFFFAOYSA-N Phosphorus Chemical compound [P] OAICVXFJPJFONN-UHFFFAOYSA-N 0.000 description 2
- 102000001253 Protein Kinase Human genes 0.000 description 2
- 241000921636 Ranunculus aquatilis Species 0.000 description 2
- 244000079489 Rorippa palustris Species 0.000 description 2
- 108050000406 Sedoheptulose-1,7-bisphosphatases Proteins 0.000 description 2
- 101100516931 Streptomyces sp. (strain CL190) nphT7 gene Proteins 0.000 description 2
- AYFVYJQAPQTCCC-UHFFFAOYSA-N Threonine Natural products CC(O)C(N)C(O)=O AYFVYJQAPQTCCC-UHFFFAOYSA-N 0.000 description 2
- 239000004473 Threonine Substances 0.000 description 2
- QIVBCDIJIAJPQS-UHFFFAOYSA-N Tryptophan Natural products C1=CC=C2C(CC(N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-UHFFFAOYSA-N 0.000 description 2
- KZSNJWFQEVHDMF-UHFFFAOYSA-N Valine Natural products CC(C)C(N)C(O)=O KZSNJWFQEVHDMF-UHFFFAOYSA-N 0.000 description 2
- 238000002835 absorbance Methods 0.000 description 2
- SYHNZXHNEJUCJR-JGWLITMVSA-N aldehydo-D-glucose 3-phosphate Chemical compound OC[C@@H](O)[C@@H](O)[C@H](OP(O)(O)=O)[C@@H](O)C=O SYHNZXHNEJUCJR-JGWLITMVSA-N 0.000 description 2
- 229940088710 antibiotic agent Drugs 0.000 description 2
- 125000003118 aryl group Chemical group 0.000 description 2
- RNBGYGVWRKECFJ-ARQDHWQXSA-N beta-D-fructofuranose 1,6-bisphosphate Chemical compound O[C@H]1[C@H](O)[C@@](O)(COP(O)(O)=O)O[C@@H]1COP(O)(O)=O RNBGYGVWRKECFJ-ARQDHWQXSA-N 0.000 description 2
- 229940004120 bifidobacterium infantis Drugs 0.000 description 2
- 239000000872 buffer Substances 0.000 description 2
- 238000011088 calibration curve Methods 0.000 description 2
- 150000001720 carbohydrates Chemical class 0.000 description 2
- 235000014633 carbohydrates Nutrition 0.000 description 2
- 238000004113 cell culture Methods 0.000 description 2
- 230000010261 cell growth Effects 0.000 description 2
- 210000000170 cell membrane Anatomy 0.000 description 2
- 230000003833 cell viability Effects 0.000 description 2
- 230000001413 cellular effect Effects 0.000 description 2
- 239000003795 chemical substances by application Substances 0.000 description 2
- 229920003211 cis-1,4-polyisoprene Polymers 0.000 description 2
- 239000002299 complementary DNA Substances 0.000 description 2
- 238000007796 conventional method Methods 0.000 description 2
- 230000007423 decrease Effects 0.000 description 2
- 238000012217 deletion Methods 0.000 description 2
- 230000037430 deletion Effects 0.000 description 2
- 108010049285 dephospho-CoA kinase Proteins 0.000 description 2
- 238000010586 diagram Methods 0.000 description 2
- 239000001177 diphosphate Substances 0.000 description 2
- 230000008034 disappearance Effects 0.000 description 2
- 230000007613 environmental effect Effects 0.000 description 2
- 238000000605 extraction Methods 0.000 description 2
- 238000004817 gas chromatography Methods 0.000 description 2
- 229940116332 glucose oxidase Drugs 0.000 description 2
- 235000019420 glucose oxidase Nutrition 0.000 description 2
- HNDVDQJCIGZPNO-UHFFFAOYSA-N histidine Natural products OC(=O)C(N)CC1=CN=CN1 HNDVDQJCIGZPNO-UHFFFAOYSA-N 0.000 description 2
- 238000009396 hybridization Methods 0.000 description 2
- 150000002430 hydrocarbons Chemical class 0.000 description 2
- 229910052738 indium Inorganic materials 0.000 description 2
- AGPKZVBTJJNPAG-UHFFFAOYSA-N isoleucine Natural products CCC(C)C(N)C(O)=O AGPKZVBTJJNPAG-UHFFFAOYSA-N 0.000 description 2
- 229960000310 isoleucine Drugs 0.000 description 2
- 239000012528 membrane Substances 0.000 description 2
- 238000010369 molecular cloning Methods 0.000 description 2
- 238000012544 monitoring process Methods 0.000 description 2
- 229930027945 nicotinamide-adenine dinucleotide Natural products 0.000 description 2
- 150000002894 organic compounds Chemical class 0.000 description 2
- 239000003208 petroleum Substances 0.000 description 2
- COLNVLDHVKWLRT-UHFFFAOYSA-N phenylalanine Natural products OC(=O)C(N)CC1=CC=CC=C1 COLNVLDHVKWLRT-UHFFFAOYSA-N 0.000 description 2
- 230000008569 process Effects 0.000 description 2
- 238000000746 purification Methods 0.000 description 2
- 238000009790 rate-determining step (RDS) Methods 0.000 description 2
- 150000003839 salts Chemical class 0.000 description 2
- 239000000243 solution Substances 0.000 description 2
- 239000007858 starting material Substances 0.000 description 2
- UCSJYZPVAKXKNQ-HZYVHMACSA-N streptomycin Chemical compound CN[C@H]1[C@H](O)[C@@H](O)[C@H](CO)O[C@H]1O[C@@H]1[C@](C=O)(O)[C@H](C)O[C@H]1O[C@@H]1[C@@H](NC(N)=N)[C@H](O)[C@@H](NC(N)=N)[C@H](O)[C@H]1O UCSJYZPVAKXKNQ-HZYVHMACSA-N 0.000 description 2
- 239000005061 synthetic rubber Substances 0.000 description 2
- 101150040618 talB gene Proteins 0.000 description 2
- 238000013518 transcription Methods 0.000 description 2
- 230000035897 transcription Effects 0.000 description 2
- LENZDBCJOHFCAS-UHFFFAOYSA-N tris Chemical compound OCC(N)(CO)CO LENZDBCJOHFCAS-UHFFFAOYSA-N 0.000 description 2
- OUYCCCASQSFEME-UHFFFAOYSA-N tyrosine Natural products OC(=O)C(N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-UHFFFAOYSA-N 0.000 description 2
- 239000004474 valine Substances 0.000 description 2
- MTCFGRXMJLQNBG-REOHCLBHSA-N (2S)-2-Amino-3-hydroxypropansäure Chemical compound OC[C@H](N)C(O)=O MTCFGRXMJLQNBG-REOHCLBHSA-N 0.000 description 1
- OWEFQTXQEHYDEJ-UHFFFAOYSA-N 2,3-dihydroxypropanal diphosphono hydrogen phosphate Chemical compound OCC(O)C=O.OP(O)(=O)OP(O)(=O)OP(O)(O)=O OWEFQTXQEHYDEJ-UHFFFAOYSA-N 0.000 description 1
- 108030005203 2-C-methyl-D-erythritol 2,4-cyclodiphosphate synthases Proteins 0.000 description 1
- 101710195531 2-C-methyl-D-erythritol 4-phosphate cytidylyltransferase, chloroplastic Proteins 0.000 description 1
- WPAMZTWLKIDIOP-UCORVYFPSA-N 2-keto-3-deoxy-L-galactonic acid Chemical compound OC[C@H](O)[C@@H](O)CC(=O)C(O)=O WPAMZTWLKIDIOP-UCORVYFPSA-N 0.000 description 1
- VJLIETBJLLTCJV-UHFFFAOYSA-N 2-methylbuta-1,3-diene Chemical compound CC(=C)C=C.CC(=C)C=C.CC(=C)C=C VJLIETBJLLTCJV-UHFFFAOYSA-N 0.000 description 1
- NPOAOTPXWNWTSH-UHFFFAOYSA-N 3-hydroxy-3-methylglutaric acid Chemical compound OC(=O)CC(O)(C)CC(O)=O NPOAOTPXWNWTSH-UHFFFAOYSA-N 0.000 description 1
- GSXSBQVSOXBYQO-XMWLYHNJSA-N 5-[2-[3-[[(2r)-4-[[[(2r,3s,4r,5r)-5-(6-aminopurin-9-yl)-4-hydroxy-3-phosphonooxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-hydroxyphosphoryl]oxy-2-hydroxy-3,3-dimethylbutanoyl]amino]propanoylamino]ethylsulfanyl]-4-methyl-5-oxopentanoic acid Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCSC(=O)C(CCC(O)=O)C)O[C@H]1N1C2=NC=NC(N)=C2N=C1 GSXSBQVSOXBYQO-XMWLYHNJSA-N 0.000 description 1
- 239000004475 Arginine Substances 0.000 description 1
- DCXYFEDJOCDNAF-UHFFFAOYSA-N Asparagine Natural products OC(=O)C(N)CC(N)=O DCXYFEDJOCDNAF-UHFFFAOYSA-N 0.000 description 1
- 241000193744 Bacillus amyloliquefaciens Species 0.000 description 1
- 241000193752 Bacillus circulans Species 0.000 description 1
- 241001328122 Bacillus clausii Species 0.000 description 1
- 241000193749 Bacillus coagulans Species 0.000 description 1
- 241000006382 Bacillus halodurans Species 0.000 description 1
- 241000193422 Bacillus lentus Species 0.000 description 1
- 241000194107 Bacillus megaterium Species 0.000 description 1
- 241000193388 Bacillus thuringiensis Species 0.000 description 1
- 239000002028 Biomass Substances 0.000 description 1
- 241000193764 Brevibacillus brevis Species 0.000 description 1
- KJQMOGOKAYDMOR-UHFFFAOYSA-N CC(=C)C=C.CC(=C)C=C Chemical compound CC(=C)C=C.CC(=C)C=C KJQMOGOKAYDMOR-UHFFFAOYSA-N 0.000 description 1
- 241000222120 Candida <Saccharomycetales> Species 0.000 description 1
- 102000004031 Carboxy-Lyases Human genes 0.000 description 1
- 108090000489 Carboxy-Lyases Proteins 0.000 description 1
- 241000195628 Chlorophyta Species 0.000 description 1
- 244000223760 Cinnamomum zeylanicum Species 0.000 description 1
- 241000272470 Circus Species 0.000 description 1
- DBPRUZCKPFOVDV-UHFFFAOYSA-N Clorprenaline hydrochloride Chemical compound O.Cl.CC(C)NCC(O)C1=CC=CC=C1Cl DBPRUZCKPFOVDV-UHFFFAOYSA-N 0.000 description 1
- 241000252867 Cupriavidus metallidurans Species 0.000 description 1
- YTBSYETUWUMLBZ-UHFFFAOYSA-N D-Erythrose Natural products OCC(O)C(O)C=O YTBSYETUWUMLBZ-UHFFFAOYSA-N 0.000 description 1
- YTBSYETUWUMLBZ-IUYQGCFVSA-N D-erythrose Chemical compound OC[C@@H](O)[C@@H](O)C=O YTBSYETUWUMLBZ-IUYQGCFVSA-N 0.000 description 1
- XPYBSIWDXQFNMH-UHFFFAOYSA-N D-fructose 1,6-bisphosphate Natural products OP(=O)(O)OCC(O)C(O)C(O)C(=O)COP(O)(O)=O XPYBSIWDXQFNMH-UHFFFAOYSA-N 0.000 description 1
- MNQZXJOMYWMBOU-VKHMYHEASA-N D-glyceraldehyde Chemical compound OC[C@@H](O)C=O MNQZXJOMYWMBOU-VKHMYHEASA-N 0.000 description 1
- 241000199914 Dinophyceae Species 0.000 description 1
- 241001481760 Erethizon dorsatum Species 0.000 description 1
- 206010056474 Erythrosis Diseases 0.000 description 1
- 241000672609 Escherichia coli BL21 Species 0.000 description 1
- 241001198387 Escherichia coli BL21(DE3) Species 0.000 description 1
- 241000195620 Euglena Species 0.000 description 1
- 241000206602 Eukaryota Species 0.000 description 1
- 241000206672 Gelidium Species 0.000 description 1
- 241000193385 Geobacillus stearothermophilus Species 0.000 description 1
- WHUUTDBJXJRKMK-UHFFFAOYSA-N Glutamic acid Natural products OC(=O)C(N)CCC(O)=O WHUUTDBJXJRKMK-UHFFFAOYSA-N 0.000 description 1
- 239000004471 Glycine Substances 0.000 description 1
- 101000729289 Homo sapiens Ribose-5-phosphate isomerase Proteins 0.000 description 1
- 238000012218 Kunkel's method Methods 0.000 description 1
- XUJNEKJLAYXESH-REOHCLBHSA-N L-Cysteine Chemical compound SC[C@H](N)C(O)=O XUJNEKJLAYXESH-REOHCLBHSA-N 0.000 description 1
- ONIBWKKTOPOVIA-BYPYZUCNSA-N L-Proline Chemical compound OC(=O)[C@@H]1CCCN1 ONIBWKKTOPOVIA-BYPYZUCNSA-N 0.000 description 1
- QNAYBMKLOCPYGJ-REOHCLBHSA-N L-alanine Chemical compound C[C@H](N)C(O)=O QNAYBMKLOCPYGJ-REOHCLBHSA-N 0.000 description 1
- ODKSFYDXXFIFQN-BYPYZUCNSA-P L-argininium(2+) Chemical compound NC(=[NH2+])NCCC[C@H]([NH3+])C(O)=O ODKSFYDXXFIFQN-BYPYZUCNSA-P 0.000 description 1
- DCXYFEDJOCDNAF-REOHCLBHSA-N L-asparagine Chemical compound OC(=O)[C@@H](N)CC(N)=O DCXYFEDJOCDNAF-REOHCLBHSA-N 0.000 description 1
- CKLJMWTZIZZHCS-REOHCLBHSA-N L-aspartic acid Chemical compound OC(=O)[C@@H](N)CC(O)=O CKLJMWTZIZZHCS-REOHCLBHSA-N 0.000 description 1
- WHUUTDBJXJRKMK-VKHMYHEASA-N L-glutamic acid Chemical compound OC(=O)[C@@H](N)CCC(O)=O WHUUTDBJXJRKMK-VKHMYHEASA-N 0.000 description 1
- ZDXPYRJPNDTMRX-VKHMYHEASA-N L-glutamine Chemical compound OC(=O)[C@@H](N)CCC(N)=O ZDXPYRJPNDTMRX-VKHMYHEASA-N 0.000 description 1
- 102000003855 L-lactate dehydrogenase Human genes 0.000 description 1
- 108700023483 L-lactate dehydrogenases Proteins 0.000 description 1
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 1
- KDXKERNSBIXSRK-YFKPBYRVSA-N L-lysine Chemical compound NCCCC[C@H](N)C(O)=O KDXKERNSBIXSRK-YFKPBYRVSA-N 0.000 description 1
- FFEARJCKVFRZRR-BYPYZUCNSA-N L-methionine Chemical compound CSCC[C@H](N)C(O)=O FFEARJCKVFRZRR-BYPYZUCNSA-N 0.000 description 1
- 241000186612 Lactobacillus sakei Species 0.000 description 1
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 description 1
- 241000186781 Listeria Species 0.000 description 1
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 description 1
- 239000004472 Lysine Substances 0.000 description 1
- FYYHWMGAXLPEAU-UHFFFAOYSA-N Magnesium Chemical compound [Mg] FYYHWMGAXLPEAU-UHFFFAOYSA-N 0.000 description 1
- 241001148031 Methanococcoides burtonii Species 0.000 description 1
- 241001340448 Mucilaginibacter Species 0.000 description 1
- 230000004988 N-glycosylation Effects 0.000 description 1
- 240000002853 Nelumbo nucifera Species 0.000 description 1
- 235000006508 Nelumbo nucifera Nutrition 0.000 description 1
- 235000006510 Nelumbo pentapetala Nutrition 0.000 description 1
- 241000192656 Nostoc Species 0.000 description 1
- 241000194109 Paenibacillus lautus Species 0.000 description 1
- 240000001090 Papaver somniferum Species 0.000 description 1
- 235000008753 Papaver somniferum Nutrition 0.000 description 1
- 241001660097 Pedobacter Species 0.000 description 1
- 102000001105 Phosphofructokinases Human genes 0.000 description 1
- 108010069341 Phosphofructokinases Proteins 0.000 description 1
- 102000004160 Phosphoric Monoester Hydrolases Human genes 0.000 description 1
- 108090000608 Phosphoric Monoester Hydrolases Proteins 0.000 description 1
- 108091000080 Phosphotransferase Proteins 0.000 description 1
- 241000235061 Pichia sp. Species 0.000 description 1
- 241000255969 Pieris brassicae Species 0.000 description 1
- ONIBWKKTOPOVIA-UHFFFAOYSA-N Proline Natural products OC(=O)C1CCCN1 ONIBWKKTOPOVIA-UHFFFAOYSA-N 0.000 description 1
- 241000168225 Pseudomonas alcaligenes Species 0.000 description 1
- 241000589774 Pseudomonas sp. Species 0.000 description 1
- 108020004511 Recombinant DNA Proteins 0.000 description 1
- 241000206572 Rhodophyta Species 0.000 description 1
- 108091028664 Ribonucleotide Proteins 0.000 description 1
- 102100031139 Ribose-5-phosphate isomerase Human genes 0.000 description 1
- 101900354897 Saccharomyces cerevisiae Mevalonate kinase Proteins 0.000 description 1
- 241000235088 Saccharomyces sp. Species 0.000 description 1
- 241000811204 Salix eriocephala Species 0.000 description 1
- 241000720795 Schizosaccharomyces sp. Species 0.000 description 1
- MTCFGRXMJLQNBG-UHFFFAOYSA-N Serine Natural products OCC(N)C(O)=O MTCFGRXMJLQNBG-UHFFFAOYSA-N 0.000 description 1
- 201000005010 Streptococcus pneumonia Diseases 0.000 description 1
- 241000193998 Streptococcus pneumoniae Species 0.000 description 1
- 241000187759 Streptomyces albus Species 0.000 description 1
- NINIDFKCEFEMDL-UHFFFAOYSA-N Sulfur Chemical compound [S] NINIDFKCEFEMDL-UHFFFAOYSA-N 0.000 description 1
- 235000016639 Syzygium aromaticum Nutrition 0.000 description 1
- 244000223014 Syzygium aromaticum Species 0.000 description 1
- 239000004098 Tetracycline Substances 0.000 description 1
- 102100028601 Transaldolase Human genes 0.000 description 1
- 241000700605 Viruses Species 0.000 description 1
- 241000607479 Yersinia pestis Species 0.000 description 1
- 244000273928 Zingiber officinale Species 0.000 description 1
- 235000006886 Zingiber officinale Nutrition 0.000 description 1
- 238000010521 absorption reaction Methods 0.000 description 1
- WDJHALXBUFZDSR-UHFFFAOYSA-M acetoacetate Chemical compound CC(=O)CC([O-])=O WDJHALXBUFZDSR-UHFFFAOYSA-M 0.000 description 1
- 230000002378 acidificating effect Effects 0.000 description 1
- 239000000654 additive Substances 0.000 description 1
- 230000000996 additive effect Effects 0.000 description 1
- 241001148470 aerobic bacillus Species 0.000 description 1
- 235000004279 alanine Nutrition 0.000 description 1
- 229910000147 aluminium phosphate Inorganic materials 0.000 description 1
- 229960000723 ampicillin Drugs 0.000 description 1
- AVKUERGKIZMTKX-NJBDSQKTSA-N ampicillin Chemical compound C1([C@@H](N)C(=O)N[C@H]2[C@H]3SC([C@@H](N3C2=O)C(O)=O)(C)C)=CC=CC=C1 AVKUERGKIZMTKX-NJBDSQKTSA-N 0.000 description 1
- 210000004102 animal cell Anatomy 0.000 description 1
- 230000000507 anthelmentic effect Effects 0.000 description 1
- ODKSFYDXXFIFQN-UHFFFAOYSA-N arginine Natural products OC(=O)C(N)CCCNC(N)=N ODKSFYDXXFIFQN-UHFFFAOYSA-N 0.000 description 1
- 235000009582 asparagine Nutrition 0.000 description 1
- 229960001230 asparagine Drugs 0.000 description 1
- 235000003704 aspartic acid Nutrition 0.000 description 1
- QVGXLLKOCUKJST-UHFFFAOYSA-N atomic oxygen Chemical compound [O] QVGXLLKOCUKJST-UHFFFAOYSA-N 0.000 description 1
- 229940097012 bacillus thuringiensis Drugs 0.000 description 1
- OQFSQFPPLPISGP-UHFFFAOYSA-N beta-carboxyaspartic acid Natural products OC(=O)C(N)C(C(O)=O)C(O)=O OQFSQFPPLPISGP-UHFFFAOYSA-N 0.000 description 1
- 230000001588 bifunctional effect Effects 0.000 description 1
- 239000004621 biodegradable polymer Substances 0.000 description 1
- 229920002988 biodegradable polymer Polymers 0.000 description 1
- QTPILKSJIOLICA-UHFFFAOYSA-N bis[hydroxy(phosphonooxy)phosphoryl] hydrogen phosphate Chemical compound OP(O)(=O)OP(O)(=O)OP(O)(=O)OP(O)(=O)OP(O)(O)=O QTPILKSJIOLICA-UHFFFAOYSA-N 0.000 description 1
- 150000001721 carbon Chemical group 0.000 description 1
- 125000004432 carbon atom Chemical group C* 0.000 description 1
- 239000001569 carbon dioxide Substances 0.000 description 1
- 229910002092 carbon dioxide Inorganic materials 0.000 description 1
- 108010031234 carbon monoxide dehydrogenase Proteins 0.000 description 1
- 230000003197 catalytic effect Effects 0.000 description 1
- 238000006555 catalytic reaction Methods 0.000 description 1
- 239000007795 chemical reaction product Substances 0.000 description 1
- 238000004587 chromatography analysis Methods 0.000 description 1
- 235000017803 cinnamon Nutrition 0.000 description 1
- 238000010960 commercial process Methods 0.000 description 1
- 230000000295 complement effect Effects 0.000 description 1
- 230000000875 corresponding effect Effects 0.000 description 1
- 239000002537 cosmetic Substances 0.000 description 1
- XUJNEKJLAYXESH-UHFFFAOYSA-N cysteine Natural products SCC(N)C(O)=O XUJNEKJLAYXESH-UHFFFAOYSA-N 0.000 description 1
- 235000018417 cysteine Nutrition 0.000 description 1
- 230000020176 deacylation Effects 0.000 description 1
- 238000005947 deacylation reaction Methods 0.000 description 1
- 239000005547 deoxyribonucleotide Substances 0.000 description 1
- 125000002637 deoxyribonucleotide group Chemical group 0.000 description 1
- 108010060155 deoxyxylulose-5-phosphate synthase Proteins 0.000 description 1
- 238000001514 detection method Methods 0.000 description 1
- 235000015872 dietary supplement Nutrition 0.000 description 1
- 230000029087 digestion Effects 0.000 description 1
- 150000004141 diterpene derivatives Chemical class 0.000 description 1
- VHJLVAABSRFDPM-QWWZWVQMSA-N dithiothreitol Chemical compound SC[C@@H](O)[C@H](O)CS VHJLVAABSRFDPM-QWWZWVQMSA-N 0.000 description 1
- 229940079593 drug Drugs 0.000 description 1
- 239000003814 drug Substances 0.000 description 1
- 229920001971 elastomer Polymers 0.000 description 1
- 239000003623 enhancer Substances 0.000 description 1
- 125000002587 enol group Chemical group 0.000 description 1
- 239000000284 extract Substances 0.000 description 1
- 239000012527 feed solution Substances 0.000 description 1
- 239000000796 flavoring agent Substances 0.000 description 1
- 235000013355 food flavoring agent Nutrition 0.000 description 1
- 238000004508 fractional distillation Methods 0.000 description 1
- RNBGYGVWRKECFJ-UHFFFAOYSA-N fructose-1,6-phosphate Natural products OC1C(O)C(O)(COP(O)(O)=O)OC1COP(O)(O)=O RNBGYGVWRKECFJ-UHFFFAOYSA-N 0.000 description 1
- 230000002068 genetic effect Effects 0.000 description 1
- 235000008397 ginger Nutrition 0.000 description 1
- 230000004153 glucose metabolism Effects 0.000 description 1
- 235000013922 glutamic acid Nutrition 0.000 description 1
- 239000004220 glutamic acid Substances 0.000 description 1
- ZDXPYRJPNDTMRX-UHFFFAOYSA-N glutamine Natural products OC(=O)C(N)CCC(N)=O ZDXPYRJPNDTMRX-UHFFFAOYSA-N 0.000 description 1
- 235000011187 glycerol Nutrition 0.000 description 1
- 229930195733 hydrocarbon Natural products 0.000 description 1
- 230000001900 immune effect Effects 0.000 description 1
- 238000010348 incorporation Methods 0.000 description 1
- 238000011534 incubation Methods 0.000 description 1
- 230000002401 inhibitory effect Effects 0.000 description 1
- 229910052816 inorganic phosphate Inorganic materials 0.000 description 1
- 238000003780 insertion Methods 0.000 description 1
- 230000037431 insertion Effects 0.000 description 1
- GSXOAOHZAIYLCY-HSUXUTPPSA-N keto-D-fructose 6-phosphate Chemical compound OCC(=O)[C@@H](O)[C@H](O)[C@H](O)COP(O)(O)=O GSXOAOHZAIYLCY-HSUXUTPPSA-N 0.000 description 1
- 150000002576 ketones Chemical class 0.000 description 1
- 229920000126 latex Polymers 0.000 description 1
- 239000004816 latex Substances 0.000 description 1
- 239000006166 lysate Substances 0.000 description 1
- 239000011777 magnesium Substances 0.000 description 1
- 229910052749 magnesium Inorganic materials 0.000 description 1
- 238000012423 maintenance Methods 0.000 description 1
- 239000000463 material Substances 0.000 description 1
- 238000005259 measurement Methods 0.000 description 1
- 230000007246 mechanism Effects 0.000 description 1
- 230000001404 mediated effect Effects 0.000 description 1
- 229960001961 meglutol Drugs 0.000 description 1
- 229930182817 methionine Natural products 0.000 description 1
- 230000002906 microbiologic effect Effects 0.000 description 1
- 239000006151 minimal media Substances 0.000 description 1
- 238000001823 molecular biology technique Methods 0.000 description 1
- 229930014626 natural product Natural products 0.000 description 1
- BOPGDPNILDQYTO-NNYOXOHSSA-N nicotinamide-adenine dinucleotide Chemical compound C1=CCC(C(=O)N)=CN1[C@H]1[C@H](O)[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OC[C@@H]2[C@H]([C@@H](O)[C@@H](O2)N2C3=NC=NC(N)=C3N=C2)O)O1 BOPGDPNILDQYTO-NNYOXOHSSA-N 0.000 description 1
- 229910052757 nitrogen Inorganic materials 0.000 description 1
- 238000003199 nucleic acid amplification method Methods 0.000 description 1
- 235000015097 nutrients Nutrition 0.000 description 1
- 238000002515 oligonucleotide synthesis Methods 0.000 description 1
- 230000003647 oxidation Effects 0.000 description 1
- 238000007254 oxidation reaction Methods 0.000 description 1
- 239000001301 oxygen Substances 0.000 description 1
- 229910052760 oxygen Inorganic materials 0.000 description 1
- 230000036961 partial effect Effects 0.000 description 1
- UXMKOCWBWVWJEG-UHFFFAOYSA-N pent-1-enyl phosphono hydrogen phosphate Chemical compound CCCC=COP(O)(=O)OP(O)(O)=O UXMKOCWBWVWJEG-UHFFFAOYSA-N 0.000 description 1
- 238000005120 petroleum cracking Methods 0.000 description 1
- 108010022393 phosphogluconate dehydratase Proteins 0.000 description 1
- 102000020233 phosphotransferase Human genes 0.000 description 1
- 238000002264 polyacrylamide gel electrophoresis Methods 0.000 description 1
- 229920001195 polyisoprene Polymers 0.000 description 1
- 229920000642 polymer Polymers 0.000 description 1
- 238000006116 polymerization reaction Methods 0.000 description 1
- 230000000379 polymerizing effect Effects 0.000 description 1
- 230000013823 prenylation Effects 0.000 description 1
- 125000002924 primary amino group Chemical group [H]N([H])* 0.000 description 1
- 235000021251 pulses Nutrition 0.000 description 1
- 238000010188 recombinant method Methods 0.000 description 1
- 230000006798 recombination Effects 0.000 description 1
- 238000007670 refining Methods 0.000 description 1
- 230000010076 replication Effects 0.000 description 1
- 108091008146 restriction endonucleases Proteins 0.000 description 1
- 239000002336 ribonucleotide Substances 0.000 description 1
- 125000002652 ribonucleotide group Chemical group 0.000 description 1
- 239000005060 rubber Substances 0.000 description 1
- 235000021309 simple sugar Nutrition 0.000 description 1
- 238000002741 site-directed mutagenesis Methods 0.000 description 1
- 239000002904 solvent Substances 0.000 description 1
- 238000002798 spectrophotometry method Methods 0.000 description 1
- 238000010186 staining Methods 0.000 description 1
- 229960005322 streptomycin Drugs 0.000 description 1
- 239000011593 sulfur Substances 0.000 description 1
- 229910052717 sulfur Inorganic materials 0.000 description 1
- 229920001059 synthetic polymer Polymers 0.000 description 1
- 235000007586 terpenes Nutrition 0.000 description 1
- 229960002180 tetracycline Drugs 0.000 description 1
- 229930101283 tetracycline Natural products 0.000 description 1
- 235000019364 tetracycline Nutrition 0.000 description 1
- 150000003522 tetracyclines Chemical class 0.000 description 1
- 235000019529 tetraterpenoid Nutrition 0.000 description 1
- 231100000331 toxic Toxicity 0.000 description 1
- 230000002588 toxic effect Effects 0.000 description 1
- 230000009466 transformation Effects 0.000 description 1
- 230000032258 transport Effects 0.000 description 1
- 230000007306 turnover Effects 0.000 description 1
- 241001148471 unidentified anaerobic bacterium Species 0.000 description 1
- 239000013598 vector Substances 0.000 description 1
- 238000005406 washing Methods 0.000 description 1
- 229910001868 water Inorganic materials 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P5/00—Preparation of hydrocarbons or halogenated hydrocarbons
- C12P5/007—Preparation of hydrocarbons or halogenated hydrocarbons containing one or more isoprene units, i.e. terpenes
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N1/00—Microorganisms, e.g. protozoa; Compositions thereof; Processes of propagating, maintaining or preserving microorganisms or compositions thereof; Processes of preparing or isolating a composition containing a microorganism; Culture media therefor
- C12N1/12—Unicellular algae; Culture media therefor
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N1/00—Microorganisms, e.g. protozoa; Compositions thereof; Processes of propagating, maintaining or preserving microorganisms or compositions thereof; Processes of preparing or isolating a composition containing a microorganism; Culture media therefor
- C12N1/14—Fungi; Culture media therefor
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N1/00—Microorganisms, e.g. protozoa; Compositions thereof; Processes of propagating, maintaining or preserving microorganisms or compositions thereof; Processes of preparing or isolating a composition containing a microorganism; Culture media therefor
- C12N1/20—Bacteria; Culture media therefor
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/70—Vectors or expression systems specially adapted for E. coli
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/10—Transferases (2.)
- C12N9/1085—Transferases (2.) transferring alkyl or aryl groups other than methyl groups (2.5)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/88—Lyases (4.)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P7/00—Preparation of oxygen-containing organic compounds
- C12P7/40—Preparation of oxygen-containing organic compounds containing a carboxyl group including Peroxycarboxylic acids
- C12P7/42—Hydroxy-carboxylic acids
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y203/00—Acyltransferases (2.3)
- C12Y203/01—Acyltransferases (2.3) transferring groups other than amino-acyl groups (2.3.1)
- C12Y203/01008—Phosphate acetyltransferase (2.3.1.8)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y401/00—Carbon-carbon lyases (4.1)
- C12Y401/02—Aldehyde-lyases (4.1.2)
- C12Y401/02009—Phosphoketolase (4.1.2.9)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y402/00—Carbon-oxygen lyases (4.2)
- C12Y402/03—Carbon-oxygen lyases (4.2) acting on phosphates (4.2.3)
- C12Y402/03027—Isoprene synthase (4.2.3.27)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y503/00—Intramolecular oxidoreductases (5.3)
- C12Y503/03—Intramolecular oxidoreductases (5.3) transposing C=C bonds (5.3.3)
- C12Y503/03002—Isopentenyl-diphosphate DELTA-isomerase (5.3.3.2)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y205/00—Transferases transferring alkyl or aryl groups, other than methyl groups (2.5)
- C12Y205/01—Transferases transferring alkyl or aryl groups, other than methyl groups (2.5) transferring alkyl or aryl groups, other than methyl groups (2.5.1)
-
- Y—GENERAL TAGGING OF NEW TECHNOLOGICAL DEVELOPMENTS; GENERAL TAGGING OF CROSS-SECTIONAL TECHNOLOGIES SPANNING OVER SEVERAL SECTIONS OF THE IPC; TECHNICAL SUBJECTS COVERED BY FORMER USPC CROSS-REFERENCE ART COLLECTIONS [XRACs] AND DIGESTS
- Y02—TECHNOLOGIES OR APPLICATIONS FOR MITIGATION OR ADAPTATION AGAINST CLIMATE CHANGE
- Y02P—CLIMATE CHANGE MITIGATION TECHNOLOGIES IN THE PRODUCTION OR PROCESSING OF GOODS
- Y02P20/00—Technologies relating to chemical industry
- Y02P20/50—Improvements relating to the production of bulk chemicals
- Y02P20/52—Improvements relating to the production of bulk chemicals using catalysts, e.g. selective catalysts
Landscapes
- Chemical & Material Sciences (AREA)
- Organic Chemistry (AREA)
- Life Sciences & Earth Sciences (AREA)
- Health & Medical Sciences (AREA)
- Engineering & Computer Science (AREA)
- Wood Science & Technology (AREA)
- Zoology (AREA)
- Genetics & Genomics (AREA)
- Bioinformatics & Cheminformatics (AREA)
- General Engineering & Computer Science (AREA)
- Biochemistry (AREA)
- General Health & Medical Sciences (AREA)
- Biotechnology (AREA)
- Microbiology (AREA)
- Biomedical Technology (AREA)
- Medicinal Chemistry (AREA)
- Molecular Biology (AREA)
- Chemical Kinetics & Catalysis (AREA)
- General Chemical & Material Sciences (AREA)
- Tropical Medicine & Parasitology (AREA)
- Virology (AREA)
- Botany (AREA)
- Mycology (AREA)
- Cell Biology (AREA)
- Physics & Mathematics (AREA)
- Biophysics (AREA)
- Plant Pathology (AREA)
- Preparation Of Compounds By Using Micro-Organisms (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Enzymes And Modification Thereof (AREA)
Description
本出願は、2011年10月7日出願の、米国特許仮出願番号第61/545,083号の優先権を主張する。この特許文献は、参照によりその全体が本明細書に組み込まれる。
37 C.F.R.§ 1.821(c)及び(e)に従い、ASCIIテキストとして提出された配列リストが、参照により本明細書に組み込まれる。テキストファイル名は、「643842004000_Sequence_Listing.txt」であり、テキストファイルの作成日は2012年10月4日であり、及びASCIIテキストファイルのサイズは102,077バイトである。
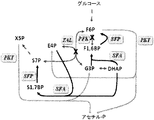
本発明は、ホスホケトラーゼポリペプチドと、メバロン酸、イソプレノイド前駆体、イソプレン、及びイソプレノイドを産生することのできるメバロン酸(MVA)経路の1種以上のポリペプチドとを含む組換え培養細胞、並びにこれらの培養細胞を含む組成物、並びにこれを産生し及び使用する方法に関する。
MVA経路のみ
1.5個グルコース+2.00個O2→1.00個イソプレン+4.00個CO2+5.00個H2O
理論収率−イソプレン0.252(g)/グルコース(g)
DXP経路
1.25個グルコース+0.50個O2→1.00個イソプレン+2.50個CO2+3.50個H2O
理論収率−イソプレン0.302(g)/グルコース(g)
MVA+ホスホケトラーゼ経路
1.22個グルコース+0.33個O2→1.00個イソプレン+2.33個CO2+3.32個H2O
理論収率−イソプレン0.309(g)/グルコース(g)
本発明の実施においては、特に断らないかぎりにおいて、当業者の技能の範囲内に含まれる従来の分子生物学(組換え技術を含む)、微生物学、細胞生物学、生化学、及び免疫学の技術を用いる。これらの手法は、次の文献:「分子クローニング:実験マニュアル(Molecular Cloning:A Laboratory Manual)」第2版(Sambrook et al.、1989年);「オリゴヌクレオチド合成(Oligonucleotide Synthesis)」(M.J.Gait,ed.,1984);「動物細胞の培養(Animal Cell Culture)」(R.I.Freshney,ed.、1987年);「酵素学的実験法(Methods in Enzymology)」(Academic Press,Inc.);「分子生物学標準プロトコル(Current Protocols in Molecular Biology)」(F.M.Ausubel et al.,eds.、1987年、定期的に改定);「PCR:ポリメラーゼ連鎖反応(PCR:The Polymerase Chain Reaction)」(Mullis et al.,eds.,1994年).Singleton et al.、「微生物学及び分子生物学辞典(Dictionary of Microbiology and Molecular Biology)第2版」、J.Wiley & Sons(New York,N.Y.1994年)、及びMarch「有機化学反応、機序及び構造第4版(Advanced Organic Chemistry Reactions,Mechanisms and Structure 4th ed.)」,John Wiley & Sons(New York,N.Y.1992)中に十分に説明されており、これらの文献は、本出願で使用される数多くの用語の一般的な指針を当業者に提供する。
用語「イソプレン」は、2−メチル−1,3−ブタジエン(CAS# 78−79−5)を指す。3,3−ジメチルアリル二リン酸(DMAPP)からピロリン酸を除去することで、揮発性のC5炭化水素を、直接的に及び最終的に生成することができる。IPP分子をDMAPP分子に結合又は重合させることは包含しない場合がある。用語「イソプレン」は、概して、本明細書に別途記載のない限り、産生方法を限定されることを意図しない。
メバロン酸依存性生合成経路(MVA経路)は、全ての高等真核生物及び特定種の細菌に存在する、重要な代謝経路である。メバロン酸経路は、タンパク質のプレニル化、細胞膜の維持、タンパク質の固定及びN−グリコシル化などの、多様な工程において使用される分子の産生に重要であり、並びにテルペン、テルペノイド、イソプレノイド、及びイソプレンの生合成時の主成分として機能するイソプレノイド前駆体分子のDMAPP及びIPPの主要な供給源を提供する。
ホスホケトラーゼ酵素は、キシルロース5−リン酸のグリセルアルデヒド3−リン酸及びアセチルリン酸への変換、並びに/又はフルクトース6−リン酸のエリトロース4−リン酸及びアセチルリン酸への変換を触媒する。特定の実施形態では、ホスホケトラーゼ酵素は、キシルロース5−リン酸のグリセルアルデヒド3−リン酸及びアセチルリン酸への変換を触媒する。他の実施形態では、ホスホケトラーゼ酵素は、フルクトース6−リン酸のエリトロース4−リン酸及びアセチルリン酸への変換を触媒する。他の実施形態では、ホスホケトラーゼポリペプチドは、セドヘプツロース−7−リン酸の生成物(例えば、リボース−5−リン酸)及びアセチルリン酸への変換を触媒する。したがって、理論に束縛されるものではないが、本明細書で説明されるとおりホスホケトラーゼを発現させることにより、炭水化物資源から生成されるアセチルリン酸量を増加させることができる。このアセチルリン酸をアセチルCoAへと変換させ、更にこれを利用して、MVA経路に関係する酵素活性により、メバロン酸、イソプレノイド前駆体分子、イソプレン及び/又はイソプレノイドを生成させることができる。したがって、炭水化物基質より生成されるこれらの化合物量を増加させることができる。あるいは、細胞内濃度の上昇という形式で生成性の向上が反映されずとも、アセチル−P及びAcCoAの生成量を増加させることができる。特定の実施形態では、細胞内アセチル−P又はアセチルCoA濃度は、ホスホケトラーゼによる反応が生じた場合でさえ変化せず一定であり、又は減少する場合すらある。
MVA経路の上流は、細胞内代謝により産生されるアセチルCo−Aを、(a)(i)チオラーゼ活性又は(ii)アセトアセチル−CoA活性、(b)HMG−CoA還元酵素、及び(c)HMG−CoAシンターゼ活性のいずれかの活性をもつポリペプチドの作用によりメバロン酸に変換する際の開始基質として利用する。最初に、チオラーゼ又はアセトアセチル−CoAシンターゼ(アセチル−CoA及びマロニル−CoAを利用する)の作用によりアセチルCo−AをアセトアセチルCoAに変換する。次に、アセトアセチル−CoAは、HMG−CoAシンターゼの酵素作用により、3−ヒドロキシ−3−メチルグルタリル−CoA(HMG−CoA)へと変換される。このCo−A誘導体をHMG−CoA還元酵素により還元してメバロン酸を生成する。この反応は、メバロン酸経路によるイソプレノイド産生の律速段階となる。
特定の実施形態では、リステリア・グレイ(L. grayi)、エンテロコッカス・フェシウム(E. faecium)、エンテロコッカス・ガリナラム(E. gallinarum)、エンテロコッカス・カセリフラブス(E. casseliflavus)及び/又はエンテロコッカス・フェカリス(E. faecalis)由来の様々なmvaE及びmvaS遺伝子を、単独で、あるいはMVA経路上流関連性タンパク質をコードしている1つ以上の他のmvaE及びmvaS遺伝子と組み合わせて選択することが、本発明の範囲内のものとして企図される。リステリア・グレイ(L. grayi)、エンテロコッカス・フェシウム(E. faecium)、エンテロコッカス・ガリナラム(E. gallinarum)、エンテロコッカス・カセリフラブス(E. casseliflavus)、及びエンテロコッカス・フェカリス(E. faecalis)では、mvaE遺伝子は、チオラーゼ及びHMG−CoA還元酵素の活性のいずれをも保有しているポリペプチドをコードする。実際に、mvaE遺伝子産物は、真性細菌で見られる、IPP生合成に関係する最初の二機能性酵素となるものであり、HMG−CoA還元酵素の第一例は、天然において他のタンパク質と融合していた(Hedl,et al.,J Bacteriol.2002 April;184(8):2116〜2122)。それに対しmvaS遺伝子は、HMG−CoAシンターゼ活性を有するポリペプチドをコードする。
mvaE遺伝子は、チオラーゼ活性及びHMG−CoA還元酵素活性を両方保有しているポリペプチドをコードする。mvaE遺伝子によりコードされているポリペプチドのチオラーゼ活性は、アセチルCo−AをアセトアセチルCoAに変換するのに対し、HMG−CoA還元酵素ポリペプチドの酵素活性は、3−ヒドロキシ−3−メチルグルタリル−CoAをメバロン酸に変換する。mvaEポリペプチド及び核酸の例としては、本明細書に記載の任意の生物資源から天然に生じるポリペプチド及び核酸、並びにmvaEポリペプチドの活性を少なくとも1つ有する、本明細書に記載の任意の生物資源から誘導されるポリペプチド変異体及び核酸変異体が挙げられる。
mvaS遺伝子は、HMG−CoAシンターゼ活性を保有するポリペプチドをコードする。このポリペプチドは、アセトアセチルCoAを、3−ヒドロキシ−3−メチルグルタリル−CoA(HMG−CoA)を変換させることができる。mvaSポリペプチド及び核酸の例としては、本明細書に記載の任意の生物資源から天然に生じるポリペプチド及び核酸、並びにmvaSポリペプチドの活性を少なくとも1つ有する、本明細書に記載の任意の生物資源から誘導されるポリペプチド変異体及び核酸変異体が挙げられる。
アセトアセチルCoAシンターゼ遺伝子(nphT7としても知られる)は、マロニルCoA及びアセチルCoAからアセトアセチルCoAを合成する活性を有し、かつ2分子のアセチルCoAからアセトアセチルCoAを合成する活性は最小限である(例えば、非活性である)酵素をコードしている遺伝子である。例えば、Okamura et al.,PNAS Vol 107,No.25,pp.11265〜11270(2010)を参照されたい。この文献中のnphT7に関する教示は、本開示に明確に組み込まれる。日本国特許公開第2008−61506(A)号及び米国特許出願公開第2010/0285549号には、放線菌のストレプトマイセス属CL190株のアセトアセチル−CoAシンターゼ遺伝子が記載されている。アセトアセチル−CoAシンターゼも、アセチルCoA:マロニルCoAアシル基転移酵素として参照され得る。使用することのできる代表的なアセトアセチル−CoAシンターゼ(又はアセチルCoA:マロニルCoAアシル基転移酵素)としては、Genbank AB540131.1が挙げられる。
本明細書に記載の組換え細胞(例えば、組換え細菌細胞)は、本明細書に記載の各種酵素経路に対し何ら操作を施していない同様の細胞を上回る量及び/又は濃度でメバロン酸を産生することができる。したがって、本明細書に記載の各種経路において調節がなされるよう遺伝子操作された組換え細胞(例えば、細菌細胞)は、メバロン酸の産生を増強させるのに有用である。
本明細書では、メバロン酸の産生方法も提供する。一部の態様では、メバロン酸の産生方法は、(a)本明細書に記載のとおりに炭素の取り込み量が増加するよう遺伝子操作された組換え細胞(上記の任意の組換え細胞)、又はメバロン酸を産生できるそれらの子孫細胞を含む、組成物を培養する工程並びに(b)メバロン酸を産生する工程、を包含する。一部の態様では、メバロン酸を産生させる方法は、メバロン酸の産生に好適な条件下で、本明細書に記載の任意の組換え細胞を培養する工程、並びに組換え細胞にメバロン酸を産生させる工程、を含む。一部の態様では、メバロン酸の産生方法は、メバロン酸を回収する工程を更に含む。
イソプレン(2−メチル−1,3−ブタジエン)は、多様な用途で使用される重要な有機化合物である。例えば、イソプレンは、合成ゴムの製造時など、数多くの化学組成物及びポリマーの合成時に、中間体又は出発物質として使用される。イソプレンはまた、多くの植物及び動物により天然に合成される重要な生体物質でもある。
本発明の一部の態様では、本明細書に記載の組成物又は方法の任意のものに記載の細胞は、メバロン酸(MVA)経路下流のポリペプチドをコードしている核酸を1つ以上更に含む。一部の態様では、MVA経路下流のポリペプチドは内在性ポリペプチドである。一部の態様では、MVA経路下流のポリペプチドをコードしている内在性核酸は、調節可能なように構成型プロモータに連結される。一部の態様では、MVA経路下流のポリペプチドをコードしている内在性核酸は、調節可能なように誘導型プロモータに連結される。一部の態様では、MVA経路下流のポリペプチドをコードしている内在性核酸は、調節可能なように高発現型プロモータに連結される。特定の態様では、細胞は、野生型細胞と比較して、内在性MVA経路下流のポリペプチドが過剰発現するよう設計する。一部の態様では、MVA経路下流のポリペプチドをコードしている内在性核酸は、調節可能なように低発現型プロモータに連結される。
本発明の一部の態様では、本明細書に記載の任意の組成物又は方法に記載の細胞(本明細書に記載されるとおりにMVA経路への炭素の取り込みを増加させるよう遺伝子操作された宿主細胞など)は、イソプレンシンターゼポリペプチド又はイソプレンシンターゼ活性を有するポリペプチドをコードしている1種以上の核酸を更に含む。一部の態様では、イソプレンシンターゼポリペプチドは内在性ポリペプチドである。一部の態様では、イソプレンシンターゼポリペプチドをコードしている内在性核酸は、構成型プロモータに調節可能なように連結される。一部の態様では、イソプレンシンターゼポリペプチドをコードしている内在性核酸は、誘導型プロモータに調節可能なように連結される。一部の態様では、イソプレンシンターゼポリペプチドをコードしている内在性核酸は、高発現型プロモータに調節可能なように連結される。特定の態様では、細胞は、野生型細胞と比較して、経路の内在性イソプレンシンターゼポリペプチドを過剰発現するよう遺伝子操作する。一部の態様では、イソプレンシンターゼポリペプチドをコードしている内在性核酸は、低発現型プロモータに調節可能なように連結される。一部の態様では、イソプレンシンターゼポリペプチドは、クズ(Pueraria)又はハコヤナギ(Populus)、又はウラジロハコヤナギとヤマナラシの交雑種(Populus alba x Populus tremula)のような交雑種に由来のポリペプチドである。
本発明の一部の態様では、本明細書に記載の任意の組成物又は方法に記載の細胞(本明細書に記載されるとおりにMVA経路への炭素の取り込みを増加させるよう遺伝子操作された宿主細胞など)は、DXSポリペプチド又はDXP経路の他のポリペプチドをコードしている1種以上の異種核酸を更に含む。一部の態様では、細胞は更に、DXSポリペプチド又は他のDXP経路ポリペプチドをコードしている内在性の核酸の染色体コピーを含む。一部の態様では、大腸菌(E. coli)細胞は更に、IDIポリペプチド及びDXSポリペプチド又は他のDXP経路ポリペプチドをコードしている核酸を1つ以上含む。一部の態様では、核酸は、イソプレンシンターゼ、IDIポリペプチド及びDXSポリペプチド又は他のDXP経路ポリペプチドをコードしている。一部の態様では、1つのプラスミドは、イソプレンシンターゼ、IDIポリペプチド及びDXSポリペプチド又は他のDXP経路ポリペプチドをコードしている。一部の態様では、複数のプラスミド(multiple plasmids)は、イソプレンシンターゼ、IDIポリペプチド及びDXSポリペプチド又は他のDXP経路ポリペプチドをコードしている。
イソプレンシンターゼ、IDI、DXP経路及び/又はMVA経路下流の核酸(及びそれらにコードされるポリペプチド)は、イソプレンシンターゼ、IDI、DXP経路及び/又はMVA経路下流の核酸を天然に含有する任意の生物から得ることができる。イソプレンは、細菌、酵母、植物及び動物などの様々な生物により天然に生成される。一部の生物は、イソプレンの産生に関係するMVA経路を含有する。イソプレンシンターゼの核酸は、例えば、イソプレンシンターゼを含有する任意の生物から得ることができる。したがってMVA経路の核酸は、例えば、MVA経路を含有する任意の生物から得ることができる。IDI及びDXP経路の核酸は、例えば、IDI及びDXP経路を含有する任意の生物から得ることができる。
本明細書に記載の組換え細胞(本明細書に記載されるとおりに炭素の取り込みを増加させるよう遺伝子操作された宿主細胞など)は、同一条件下で培養した場合に、ホスホケトラーゼポリペプチドの異種核酸の1種以上のコピー、MVA経路のポリペプチドをコードしている異種核酸の1種以上のコピー、及びイソプレンシンターゼポリペプチドをコードしている1種以上の異種核酸を含まない同様の細胞と比較して、高濃度でイソプレンを産生することができる。細胞には、IDIポリペプチドをコードしている1種以上の異種核酸を更に含ませてもよい。特定の実施形態では、ホスホケトラーゼポリペプチドは、ラクトバチルス・ロイテリ(Lactobacillus reuteri)、ビフィドバクテリウム・ロンガム(Bifidobacterium longum)、フェリモナス・バレアリカ(Ferrimonas balearica)、ペドバクター・サルタンス(Pedobactor saltans)、ストレプトマイセス・グリセウス(Streptomyces griseus)、及び/又はノカルジオプシス・ダッソンビレイ(Nocardiopsis dassonvillei)に由来する。一実施形態では、組換え細胞は、ラクトバチルス・ロイテリ(Lactobacillus reuteri)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。別の実施形態では、組換え細胞は、ビフィドバクテリウム・ロングム(Bifidobacterium longum)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。更に別の実施形態では、組換え細胞は、フェリモナス・バレアリカ(Ferrimonas balearica)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。更に他の実施形態では、組換え細胞は、ペドバクター・サルタンス(Pedobactor saltans)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。別の実施形態では、組換え細胞は、ストレプトマイセス・グリセウス(Streptomyces griseus)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。更に別の実施形態では、組換え細胞は、ノカルジオプシス・ダッソンビエイ(Nocardiopsis dassonvillei)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。他の実施形態では、本明細書に記載の組換え細胞は、クロストリジウム・アセトブチリカム(Clostridium acetobutylicum)、ラクトバチルス・ロイテリ(Lactobacillus reuteri)、ラクトバチルス・プランタラム(Lactobacillus plantarum)、ラクトバチルス・パラプランタルム(Lactobacillus paraplantarum)、ビフィドバクテリウム・ロンガム(Bifidobacterium longum)、ビフィドバクテリウム・アニマリス(Bifidobacterium animalis)、ビフィドバクテリウム・ブレビ(Bifidobacterium breve)、エンテロコッカス・ガリナラム(Enterococcus gallinarum)、ガードネレラ・バギナリス(Gardnerella vaginalis)、フェリモナス・バレアリカ(Ferrimonas balearica)、ムチラギニバクター・パルジス(Mucilaginibacter paludis)、ノストック・パンクチホルム(Nostoc punctiforme)、ノストック・パンクチホルム(Nostoc punctiforme)PCC 73102、パントエア(Pantoea)、ペドバクター・サルタンス(Pedobactor saltans)、ラーネラ・アクアティリス(Rahnella aquatilis)、ロードシュードモナス・パルストリス(Rhodopseudomonas palustris)、ストレプトマイセス・グリセウス(Streptomyces griseus)、ストレプトマイセス・アベルミティリス(Streptomyces avermitilis)、ノカルジオプシス・ダッソンビレイ(Nocardiopsis dassonvillei)、及び/又はサーモビフィダ・フスカ(Thermobifida fusca)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。更に他の実施形態では、本明細書に記載の組換え細胞は、クロストリジウム・アセトブチリカム(Clostridium acetobutylicum)、ビフィドバクテリウム・ロンガム(Bifidobacterium longum)、及び/又はエンテロコッカス・ガリナラム(Enterococcus gallinarum)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。一態様では、組換え細胞は、クロストリジウム・アセトブチリカム(Clostridium acetobutylicum)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。別の実施形態では、組換え細胞は、ビフィドバクテリウム・ロングム(Bifidobacterium longum)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。更に別の実施形態では、組換え細胞は、エンテロコッカス・ガリナラム(Enterococcus gallinarum)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。
本開示では、本明細書に記載の任意の組換え細胞を培養する工程を含む、イソプレンの産生方法も提供される。一態様では、イソプレンは、ホスホケトラーゼポリペプチド、MVA経路のポリペプチドを1種以上、及びイソプレンシンターゼポリペプチドをコードしている、1種以上の異種核酸を含む、組換え細胞を培養することにより産生することができる。特定の実施形態では、ホスホケトラーゼポリペプチドは、ラクトバチルス・ロイテリ(Lactobacillus reuteri)、ビフィドバクテリウム・ロンガム(Bifidobacterium longum)、フェリモナス・バレアリカ(Ferrimonas balearica)、ペドバクター・サルタンス(Pedobactor saltans)、ストレプトマイセス・グリセウス(Streptomyces griseus)、及び/又はノカルジオプシス・ダッソンビレイ(Nocardiopsis dassonvillei)に由来する。一実施形態では、組換え細胞は、ラクトバチルス・ロイテリ(Lactobacillus reuteri)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。別の実施形態では、組換え細胞は、ビフィドバクテリウム・ロングム(Bifidobacterium longum)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。更に別の実施形態では、組換え細胞は、フェリモナス・バレアリカ(Ferrimonas balearica)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。更に他の実施形態では、組換え細胞は、ペドバクター・サルタンス(Pedobactor saltans)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。別の実施形態では、組換え細胞は、ストレプトマイセス・グリセウス(Streptomyces griseus)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。更に別の実施形態では、組換え細胞は、ノカルジオプシス・ダッソンビエイ(Nocardiopsis dassonvillei)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。他の実施形態では、本明細書に記載の組換え細胞は、クロストリジウム・アセトブチリカム(Clostridium acetobutylicum)、ラクトバチルス・ロイテリ(Lactobacillus reuteri)、ラクトバチルス・プランタラム(Lactobacillus plantarum)、ラクトバチルス・パラプランタルム(Lactobacillus paraplantarum)、ビフィドバクテリウム・ロンガム(Bifidobacterium longum)、ビフィドバクテリウム・アニマリス(Bifidobacterium animalis)、ビフィドバクテリウム・ブレビ(Bifidobacterium breve)、エンテロコッカス・ガリナラム(Enterococcus gallinarum)、ガードネレラ・バギナリス(Gardnerella vaginalis)、フェリモナス・バレアリカ(Ferrimonas balearica)、ムチラギニバクター・パルジス(Mucilaginibacter paludis)、ノストック・パンクチホルム(Nostoc punctiforme)、ノストック・パンクチホルム(Nostoc punctiforme)PCC 73102、パントエア(Pantoea)、ペドバクター・サルタンス(Pedobactor saltans)、ラーネラ・アクアティリス(Rahnella aquatilis)、ロードシュードモナス・パルストリス(Rhodopseudomonas palustris)、ストレプトマイセス・グリセウス(Streptomyces griseus)、ストレプトマイセス・アベルミティリス(Streptomyces avermitilis)、ノカルジオプシス・ダッソンビレイ(Nocardiopsis dassonvillei)、及び/又はサーモビフィダ・フスカ(Thermobifida fusca)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。更に他の実施形態では、本明細書に記載の組換え細胞は、クロストリジウム・アセトブチリカム(Clostridium acetobutylicum)、ビフィドバクテリウム・ロンガム(Bifidobacterium longum)、及び/又はエンテロコッカス・ガリナラム(Enterococcus gallinarum)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。一態様では、組換え細胞は、クロストリジウム・アセトブチリカム(Clostridium acetobutylicum)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。別の実施形態では、組換え細胞は、ビフィドバクテリウム・ロングム(Bifidobacterium longum)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。更に別の実施形態では、組換え細胞は、エンテロコッカス・ガリナラム(Enterococcus gallinarum)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。
イソプレノイドは、多くの生物において、MVA経路の最終産物であるイソプレノイド前駆体分子の合成によりとができる。上記のように、イソプレノイドは、重要な部類の化合物として存在し、例えば、食品及び飼料用サプリメント、風味及び臭気化合物、並びに抗癌、抗マラリア、抗真菌及び抗菌化合物を含む。
本発明の組換え細胞は、イソプレノイド及びイソプレノイド前駆体分子DMAPP及びIPPの産生量を増加させる能力がある。イソプレノイドの例としては、限定するものではないが、ヘミテルペノイド、モノテルペノイド、セスキテルペノイド、ジテルペノイド、セステルテルペノイド、トリテルペノイド、テトラテルペノイド、及びより高分子量のポリテルペノイドが挙げられる。一部の態様では、ヘミテルペノイドは、プレノール(すなわち、3−メチル−2−ブテン−1−オール)、イソプレノール(すなわち、3−メチル−3−ブテン−1−オール)、2−メチル−3−ブテン−2−オール、又はイソ吉草酸である。一部の態様では、モノテルペノイドは、限定するものではないが、ゲラニルピロリン酸、オイカリプトール、リモネン、又はピネンであってよい。一部の態様では、セスキテルペノイドはファルネシルピロリン酸、アルテミシニン、又はビサボロールである。一部の態様では、ジテルペノイドは、限定するものではないが、ゲラニルゲラニルピロリン酸、レチノール、レチナール、フィトール、タキソール、フォルスコリン、又はアフィジコリンであってよい。一部の態様では、トリテルペノイドは、スクアレン又はラノステロールであってよい。イソプレノイドは、アビエタジエン、アモルファジエン、カレン、α−ファルネセン(α-famesene)、β−ファルネセン、ファルネソール、ゲラニオール、ゲラニルゲラニオール、リナロール、リモネン、ミルセン、ネロリドール、オシメン、パチョロール、β−ピネン、サビネン、γ−テルピネン、テルピンデン(terpindene)及びバレンセンからなる群から選択することもできる。
本発明の一部の態様では、本明細書において任意の組成物又は方法に記載されるように、細胞は、更に、上記の通りにメバロン酸(MVA)経路のポリペプチドを1種以上コードしている核酸、並びにポリプレニルピロリン酸シンターゼポリペプチドをコードしている1種以上の核酸を更に含む。ポリプレニルピロリン酸シンターゼポリペプチドは、内在性ポリペプチドであってよい。ポリプレニルピロリン酸シンターゼポリペプチドをコードしている内在性核酸は、構成型プロモータに調節可能なように連結させてよく、あるいは同様に、調節可能なように誘導型プロモータに連結させてもよい。ポリプレニルピロリン酸シンターゼポリペプチドをコードしている内在性核酸は、更に、調節可能なように高発現型プロモータと連結させてもよい。あるいは、ポリプレニルピロリン酸シンターゼポリペプチドをコードしている内在性の核酸は、調節可能なように低発現型プロモータと連結させてもよい。詳細には、細胞は、野生型細胞と比較して、内在性のポリプレニルピロリン酸シンターゼポリペプチドを過剰発現するよう遺伝子操作することができる。
本明細書に記載の組換え細胞(例えば、組換え細菌細胞)は、同一条件下で培養した場合に、ホスホケトラーゼをコードしている異種核酸の1種以上のコピー、MVA経路のポリペプチドをコードしている異種核酸の1種以上のコピー、及びポリプレニルピロリン酸シンターゼポリペプチドをコードしている1種以上の異種核酸を含まない同様の細胞と比較して、多量に及び/又は高濃度にイソプレノイド前駆体及び/又はイソプレノイドを産生することができる。特定の実施形態では、ホスホケトラーゼペプチドをコードしている異種核酸の1種以上のコピーは、ラクトバチルス・ロイテリ(Lactobacillus reuteri)、ビフィドバクテリウム・ロングム(Bifidobacterium longum)、フェリモナス・バレアリカ(Ferrimonas balearica)、ペドバクター・サルタンス(Pedobactor saltans)、ストレプトマイセス・グリセウス(Streptomyces griseus)、及び/又はノカルジオプシス・ダッソンビエイ(Nocardiopsis dassonvillei)に由来するものである。一実施形態では、組換え細胞は、ラクトバチルス・ロイテリ(Lactobacillus reuteri)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。別の実施形態では、組換え細胞は、ビフィドバクテリウム・ロングム(Bifidobacterium longum)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。更に別の実施形態では、組換え細胞は、フェリモナス・バレアリカ(Ferrimonas balearica)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。更に他の実施形態では、組換え細胞は、ペドバクター・サルタンス(Pedobactor saltans)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。別の実施形態では、組換え細胞は、ストレプトマイセス・グリセウス(Streptomyces griseus)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。更に別の実施形態では、組換え細胞は、ノカルジオプシス・ダッソンビエイ(Nocardiopsis dassonvillei)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。他の実施形態では、本明細書に記載の組換え細胞は、クロストリジウム・アセトブチリカム(Clostridium acetobutylicum)、ラクトバチルス・ロイテリ(Lactobacillus reuteri)、ラクトバチルス・プランタラム(Lactobacillus plantarum)、ラクトバチルス・パラプランタルム(Lactobacillus paraplantarum)、ビフィドバクテリウム・ロンガム(Bifidobacterium longum)、ビフィドバクテリウム・アニマリス(Bifidobacterium animalis)、ビフィドバクテリウム・ブレビ(Bifidobacterium breve)、エンテロコッカス・ガリナラム(Enterococcus gallinarum)、ガードネレラ・バギナリス(Gardnerella vaginalis)、フェリモナス・バレアリカ(Ferrimonas balearica)、ムチラギニバクター・パルジス(Mucilaginibacter paludis)、ノストック・パンクチホルム(Nostoc punctiforme)、ノストック・パンクチホルム(Nostoc punctiforme)PCC 73102、パントエア(Pantoea)、ペドバクター・サルタンス(Pedobactor saltans)、ラーネラ・アクアティリス(Rahnella aquatilis)、ロードシュードモナス・パルストリス(Rhodopseudomonas palustris)、ストレプトマイセス・グリセウス(Streptomyces griseus)、ストレプトマイセス・アベルミティリス(Streptomyces avermitilis)、ノカルジオプシス・ダッソンビレイ(Nocardiopsis dassonvillei)、及び/又はサーモビフィダ・フスカ(Thermobifida fusca)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。更に他の実施形態では、本明細書に記載の組換え細胞は、クロストリジウム・アセトブチリカム(Clostridium acetobutylicum)、ビフィドバクテリウム・ロンガム(Bifidobacterium longum)、及び/又はエンテロコッカス・ガリナラム(Enterococcus gallinarum)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。一態様では、組換え細胞は、クロストリジウム・アセトブチリカム(Clostridium acetobutylicum)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。別の実施形態では、組換え細胞は、ビフィドバクテリウム・ロングム(Bifidobacterium longum)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。更に別の実施形態では、組換え細胞は、エンテロコッカス・ガリナラム(Enterococcus gallinarum)から単離されたホスホケトラーゼをコードしている異種核酸の1種以上のコピーを含む。一部の態様では、ホスホケトラーゼをコードしている異種核酸の1種以上のコピー、MVA経路のポリペプチドをコードしている異種核酸の1種以上のコピー、及びポリプレニルピロリン酸シンターゼポリペプチドをコードしている1種以上の異種核酸は、宿主細胞の染色体に組み込まれた異種核酸である。組換え細胞は、ホスホケトラーゼポリペプチドを含まないイソプレノイド及び/又はイソプレノイド前駆体産生組換え細胞と比較した場合に、少なくとも5%多量のイソプレノイド前駆体及び/又はイソプレノイドを産生することができる。あるいは、組換え細胞は、ホスホケトラーゼをコードしている1種以上の異種核酸を発現しないイソプレノイド及び/又はイソプレノイド前駆体産生細胞によるイソプレノイド及び/又はイソプレノイド前駆体の産生と比較して、1%、2%、3%、4%、5%、6%、7%、8%、9%、10%、11%、12%、13%、14%、又は15%超並びにこれらの数字の間の任意の数値だけ多量にイソプレノイド前駆体及び/又はイソプレノイドを産生することができる。本明細書に記載の特定の実施形態では、本明細書に記載の方法は、MVA産生に取り込まれる炭素を増加させることにより、ホスホケトラーゼペプチドをコードしている1種以上の異種核酸を発現せずかつメバロン酸産生に取り込まれる炭素を増加させるよう遺伝子操作されていないイソプレノイド及び/又はイソプレノイド前駆体産生細胞によるイソプレノイド及び/又はイソプレノイド前駆体の産生と比較して、イソプレノイド及び/又はイソプレノイド前駆体の産生を増強させる遺伝子操作を更に行った宿主細胞を含む。
本明細書では、イソプレノイド前駆体分子及び/又はイソプレノイドの産生方法も提供され、方法は、ホスホケトラーゼ及びポリプレニルピロリン酸シンターゼポリペプチドをコードしている1種以上の異種核酸を含む組換え細胞(例えば、組換え細菌細胞)を培養することを含む。特定の実施形態では、組換え細胞は、MVA経路上流のポリペプチド及びMVA経路下流のポリペプチドをコードしている1種以上の異種核酸を更に含む。イソプレノイド前駆体分子及び/又はイソプレノイドは、本明細書に記載の任意の細胞から、及び本開示の任意の方法に従って、産生することができる。グルコースなどの六炭糖といった炭水化物から、イソプレノイド前駆体分子及び/又はイソプレノイドを産生する目的で、任意の細胞を使用することができる。
本明細書に記載の任意の組成物及び方法には好適なベクターを使用することができる。例えば、適切なベクターを使用して、ホスホケトラーゼ、MVA経路上流のポリペプチド(限定するものではないが、mvaE及びmvaSポリペプチド)、MVA経路下流のポリペプチド、イソプレンシンターゼ、又はポリプレニルピロリン酸シンターゼをコードしている遺伝子の1つ以上のコピーの、特定の宿主細胞(例えば、大腸菌(E. coli))における発現を最適化することができる。一部の態様では、ベクターには選択マーカーを含有させる。選択可能なマーカーの例としては、これらに限定されるものではないが、抗生物質耐性核酸(例えば、カナマイシン、アンピシリン、カルベニシリン、ゲンタマイシン、ヒグロマイシン、フェロマイシン、ブレオマイシン、ネオマイシン又はクロラムフェニコール)、及び/又はホスト細胞に栄養的な利点などの代謝的な利点を与える核酸が挙げられる。一部の態様では、選択マーカーは用いずに、ホスホケトラーゼ、MVA経路上流のポリペプチド(限定するものではないが、mvaE及びmvaSポリペプチド)、MVA経路下流のポリペプチド、リステリア・グレイ(L. grayi)、エンテロコッカス・フェシウム(E. faecium)、エンテロコッカス・ガリナラム(E. gallinarum)、エンテロコッカス・カッセリフラバス(E. casseliflavus)、及び/又はエンテロコッカス・フェカリス(E. faecalis)由来のmvaE及びmvaS核酸、イソプレンシンターゼ、又はポリプレニルピロリン酸シンターゼの核酸の1つ以上のコピーを宿主細胞のゲノムに組み込むことができる。
好適な技術を使用して、ホスホケトラーゼ、MVA経路上流のポリペプチド(限定するものではないが、mvaE及びmvaSポリペプチドなど)、MVA経路下流のポリペプチド、及び/又はMVA経路下流のポリペプチドの1つ以上のコピーをコードしている核酸を細胞に導入することができる。更に、イソプレンシンターゼ、IDI、DXP経路及び/又はポリプレニルピロリン酸シンターゼの核酸、又はこれらを含むベクターは、形質転換、電気穿孔法、核マイクロインジェクション、形質導入、形質移入(例えばリポフェクション介在型若しくはDEAE−デキストラン介在型トランスフェクション、又は組換えファージウイルスを使用した形質移入)、リン酸カルシウムDNA沈殿物とのインキュベーション、DNAコーティングした微粒子を用いる高速遺伝子銃照射、及びプロトプラスト融合などの、宿主細胞内にDNAコンストラクト又はベクターを導入するための標準的な技術を使用して宿主細胞(例えば本明細書において述べられるような植物細胞、真菌細胞、酵母細胞、又は細菌細胞)内に挿入することができる。一般的な形質転換法は、当該技術分野で既知である(例えば、分子生物学領域の現行のプロトコル(F.M.Ausubel et al.(eds.)Chapter 9,1987;Sambrook et al.,「分子クローニング(Molecular Cloning)」:「実験室マニュアル(A Laboratory Manual)第2版」,Cold Spring Harbor,1989;及びCampbell et al.,Curr.Genet.16:53〜56,1989を参照されたい)。導入された核酸は、染色体DNAに組み込むことができ、又は染色体外の複製配列として維持することができる。形質転換体は、当該技術分野において既知の任意の方法により選別することができる。形質転換体の選別に好適な方法は、国際公開第2009/076676号、米国特許出願公開第2009/0203102号、国際公開第2010/003007号、米国特許出願公開第2010/0048964号、国際公開第2009/132220号、及び米国特許出願公開第2010/0003716に記載される。
当業者であれば、具体的な宿主株における遺伝子発現を最適化する特定の配列を含有するよう、発現ベクターが設計されることを認識するであろう。このような最適化配列としては、限定するものではないが、複製起点、プロモータ、及びエンハンサが挙げられる。本明細書で参照するベクター及び配列は例示目的で記載され、本発明の範囲を狭めることを意味するものではない。
クエン酸シンターゼによる経路
クエン酸シンターゼは、オキサロ酢酸とアセチルCoAを縮合させることによるクエン酸(トリカルボン酸(TCA)回路の代謝生成物)の生成を触媒する(Ner,S.et al.1983.Biochemistry 22,:5243〜5249;Bhayana,V.and Duckworth,H.1984.Biochemistry 23:2900〜2905)。大腸菌(E. coli)では、gltAによりコードされたこの酵素は、二量体サブユニットからなる三量体様の挙動を示す。六量体の形成により、酵素はNADHによりアロステリックに制御されるようになる。この酵素は、これまでに広く研究されている(Wiegand,G.,and Remington,S.1986.Annual Rev.Biophysics Biophys.Chem.15:97〜117;Duckworth et al.1987.Biochem Soc Symp.54:83〜92;Stockell,D.et al.2003.J.Biol.Chem.278:35435〜43;Maurus,R.et al.2003.Biochemistry.42:5555〜5565)。NADHによるアロステリック阻害を回避するにあたって、これまでに、バチルス・サブチリス(Bacillus subtilis)NADH非感受性クエン酸シンターゼによる置き換え、又はこの酵素の添加が検討されている(Underwood et al.2002.Appl.Environ.Microbiol.68:1071〜1081;Sanchez et al.2005.Met.Eng.7:229〜239)。
ホスホトランスアセチラーゼ((大腸菌(E. coli)において(i)pta(Shimizu et al.1969.Biochim.Biophys.Acta 191:550〜558又は(ii)eutD(Bologna et al.2010.J of Microbiology.48:629〜636によりコードされている)は、アセチル−CoA及びアセチルリン酸(アセチル−P)の可逆的な変換を触媒するのに対し、酢酸キナーゼ(大腸菌(E. coli)においてackAによりコードされる)(Kakuda,H.et al.1994.J.Biochem.11:916〜922)はアセチル−Pを使用して酢酸を生成する。これらの遺伝子は、大腸菌においてオペロンとして転写され得る。これらの遺伝子は共に、ATPの放出により酢酸の異化を触媒する。したがって、ホスホトランスアセチラーゼの活性を増強させることにより、アセチル−CoAに変換されるアセチル−Pの量を増加させることができる。特定の実施形態では、増強は、染色体中の遺伝子上流に上方調節されたプロモータを配置するか、又はプラスミドにおいて適切なプロモータの下流に遺伝子のコピーを配置するかによりなすことができる。酢酸に変換されるアセチルCoAの量を減少させる目的で、酢酸キナーゼ遺伝子(例えば、内在性酢酸キナーゼ遺伝子)の活性を減少又は減弱させることができる。特定の実施形態では、減弱は、酢酸キナーゼ(ackA)を欠失させることにより実施される。このような欠失は、遺伝子をクロラムフェニコールカセットにより置き換えた後、カセットを除去させることにより実施される。一部の態様では、酢酸キナーゼの活性は、内在性酢酸キナーゼの活性を減少させることにより調節される。このような調節は、内在性酢酸キナーゼ遺伝子のプロモータを常時低発現型合成プロモータと置き換えることにより行うことができる。特定の実施形態では、このような酢酸キナーゼ遺伝子の減弱は、ホスホトランスアセチラーゼ(pta)遺伝子の発現を干渉することによりなされるべきである。酢酸は多様な理由により大腸菌(E. coli)により生成される(Wolfe,A.2005.Microb.Mol.Biol.Rev.69:12〜50)。理論に束縛されるものではないが、ackAを欠失させることにより、炭素の酢酸の産生への転用が減少し(ackAはアセチル−CoAを利用するため)、メバロン酸、イソプレノイド前駆体、イソプレン及び/又はイソプレノイドの収率が増加することになる。
大腸菌(E. coli)では、D−乳酸は、乳酸デヒドロゲナーゼ(ldhAによりコード:図1)により、ピルビン酸から生成される(Bunch,P.et al.1997.Microbiol.143:187〜195)。乳酸の生成はNADHの酸化によりなされるため、乳酸は、酸素制限下で、かつ還元当量のすべてを収容できない場合に生成されることになる。したがって、乳酸の生成は、炭素消費の原因となり得る。そのため、メバロン酸産生(並びに必要に応じてイソプレン、イソプレノイド前駆体及びイソプレノイドの産生)への炭素の取り込みを向上させるため、当業者は、酵素活性を低下させるなどして乳酸デヒドロゲナーゼの活性を調節することができる。
グリセルアルデヒド3−リン酸脱水素酵素(gapA及び/又はgapB)は、解糖に重要な酵素であり、グリセルアルデヒド3−リン酸の1,3−ビホスホ−D−グリセリン酸への変換を触媒する(Branlant G.and Branlant C.1985.Eur.J.Biochem.150:61〜66)。
エントナー・ドゥドロフ(ED)経路は、エムデン・マイヤーホフ・パルナス(EMP−解糖)経路とは異なる経路である。大腸菌(E. coli)などの一部の生物がED経路及びEMP経路の両方を内包するのに対し、その他の生物はいずれか1つの経路のみを有する。バチルス・サブチリス(Bacillus subtilis)は、EMP経路のみを有するのに対し、ザイモモナス・モビリス(Zymomonas mobilis)はED経路のみを有する(Peekhaus and Conway.1998.J.Bact.180:3495〜3502;Stulke and Hillen.2000.Annu.Rev.Microbiol.54,849〜880;Dawes et al.1966.Biochem.J.98:795〜803)。フルクトース二リン酸アルドラーゼ(fba、fbaA、fbaB、及び/又はfbaC)はエントナー・ドゥドロフ経路と相互作用し、フルクトース1,6−2リン酸のジヒドロキシアセトンリン酸(DHAP)及びグリセルアルデヒド3−リン酸(GAP)への変換を可逆的に触媒する(Baldwin S.A.,et.al.,Biochem J.(1978)169(3):633〜41)。
大腸菌(E. coli)は、ペントースリン酸経路を使用してヘキソース及びペントースを分解し、各種代謝経路に関係する中間体を細胞に提供する。この経路は、NADPHの主要な生成経路でもある。ペントースリン酸経路は、酸化経路(グルコース6−リン酸1−脱水素酵素(zwf)、6−ホスホグルコノラクトナーゼ(pgl)又は6−ホスホグルコネート脱水素酵素(gnd)などの酵素による)及び非酸化経路(トランスケトラーゼ(tktA及び/又はtktB)、トランスアルドラーゼ(talA又はtalB)、リボース−5−リン酸−エピメラーゼ及び(又は)リボース−5−リン酸エピメラーゼ、リブロース−5−リン酸イソメラーゼ(rpiA及び/又はrpiB)及び/又はリブロース−5−リン酸3−エピメラーゼ(rpe)などの酵素による)から構成される(Sprenger.1995.Arch.Microbiol.164:324〜330)。
ホスホフルクトキナーゼは、解糖系でフルクトース6−リン酸のリン酸化を触媒する重要な酵素である。大腸菌(E. coli)はpfkA及びpfkBによりコードされる2種のイソ酵素を有する。細胞におけるホスホフルクトキナーゼ活性の大部分はpfkAによるものである(Kotlarz et al.1975,Biochim.Biophys.Acta,381:257〜268)。
ピルビン酸のアセチル−CoAへの脱炭酸を触媒するピルビン酸脱水素酵素複合体は、遺伝子aceE、aceF、及びlpdAによりコードされるタンパク質から構成される。これらの遺伝子の転写は、複数の制御因子により制御される。したがって、当業者は、ピルビン酸脱水素酵素複合体の活性を調節して、アセチル−CoAを増加することができる。調節により、ピルビン酸脱水素酵素複合体の活性及び/又は発現(例えば、恒常的発現)を増加させることができる。このような調節は、異なる手法により、例えば、PL.6(aattcatataaaaaacatacagataaccatctgcggtgataaattatctctggcggtgttgacataaataccactggcggtgatactgagcacatcagcaggacgcactgaccaccatgaaggtg−ラムダプロモーター、GenBank NC_001416(配列番号14))などの強力な構成型プロモータをオペロンの前に配置することにより、あるいは構成的に(恒常的に)発現する合成プロモータを1種以上用いることにより行うことができる。
ホスホエノールピルビン酸依存型ホスホトランスフェラーゼ系(PTS)は、工程において、膜を介した糖基質の輸送及びリン酸化を同時に行う多成分系であり、解糖中間体のホスホエノールピルビン酸(PEP)によるエネルギー供給に依存する系である。PTSを調節する遺伝子の大部分はオペロンとしてクラスター化している。例えば、大腸菌(E. coli)のptsオペロン(ptsHIcrr)は、ホスホエノールピルビン酸依存型ホスホトランスフェラーゼ系(PTS)に中心的な3種類のタンパク質、すなわちHPr(ptsH)、酵素I(ptsI)及びEIIIGlc(crr)タンパク質をコードしているptsH、ptsI、及びcrr遺伝子から構成される。これら3つの遺伝子は複合型オペロンとして構成され、末端遺伝子crrの発現の大部分は、ptsI内のプロモータ領域から開始される。ptsオペロンの遺伝子の他、ptsGは、ホスホトランスフェラーゼ系のグルコース特異的な輸送体ptsGをコードしている。このプロモータ領域から開始される転写は、カタボライト活性化タンパク質(CAP)−環状AMP(cAMP)の正の調節下にあり、グルコース(PTS基質)存在下での生育中に増強される。更に、ppsA遺伝子は、ホスホトランスフェラーゼ系(PTS)の活性に必要とされるホスホエノールピルビン酸(PEP)の産生に関係するホスホエノールピルビン酸シンターゼをコードする。ピルビン酸脱水素酵素経路又はPTS経路を介し、ホスホエノールピルビン酸シンターゼにより炭素の取り込みを調整する。Postma,P.W.,et al.,Microbiol Rev.(1993),57(3):543〜94)を参照されたい。この非特許文献は、参照によりその全体が本明細書に組み込まれる。
本明細書に記載の特定の実施形態では、植物バイオマス由来の糖を、メバロン酸など、イソプレノイド前駆体、イソプレン及び/又はイソプレノイドなどの所望の産物へと変換するにあたって、キシロースを利用することが望ましい。一部の生物では、キシロースを利用するにあたり、代謝用のフルクトース−6−リン酸に変換する際に、ペントースリン酸経路を使用する必要がある。生物は、グルコースによる異化代謝抑制を不活性化させることにより、あるいは他の生物において見られるキシロースオペロン由来の遺伝子を異種発現させることにより、キシロースの利用効率を増強させるよう遺伝子操作することができる。以下に記載されるとおりにキシロース経路を遺伝子操作することで、ホスホケトラーゼ経路によるメバロン酸、イソプレノイド前駆体、イソプレン及び/又はイソプレノイドの産生を増強させることができる。
嫌気的に又はヘテロ発酵を行い生育することのできる一部の微生物は、解糖経路の代わりに、あるいは解糖経路に加えて、ホスホケトラーゼ経路を包含する。この経路は、ペントースリン酸経路の酵素、トランスアルドラーゼ及びトランスケトラーゼの活性に依存する。したがって、本明細書に記載のとおりにホスホケトラーゼ酵素を有するよう遺伝子操作した宿主細胞を、内在性又は異種のいずれかの形態のトランスケトラーゼ及びトランスアルドラーゼを過剰発現して、経路の取り込みを増加させ、毒性中間体の濃度を減少させ、非産生的経路への中間体の迂回を減少させ、並びに総収率並びにメバロン酸、イソプレノイド前駆体、イソプレン及び/又はイソプレノイド産生を向上させるよう更に遺伝子操作することができる。
本明細書に記載の任意の酵素及び/又は酵素経路、本明細書に記載の任意の組み合わせ(2、3、4、5、6、7、8、9、10、11、12、13、又は14)の酵素及び/又は酵素経路を調節する分子操作が明らかに企図されることは理解されたい。組み合わせての記載を容易にするため、クエン酸シンターゼ(gltA)はAとして表記し、ホスホトランスアセチラーゼ(pta)はBとして表記し、酢酸キナーゼ(ackA)はCとして表記し、乳酸脱水素酵素(ldhA)はDとして表記し、グリセルアルデヒド3−リン酸脱水素酵素(gap)はEとして表記し、ピルビン酸脱炭酸酵素(aceE、aceF、及び/又はlpdA)はFとして表記し、ホスホグルコン酸デヒドラターゼ(edd)はGとして表記し、2−ケト−3−デオキシグルコン酸6−リン酸アルドラーゼ(eda)はHとして表記し、ホスホフルクトキナーゼはIとして表記し、トランスアルドラーゼはJとして表記し、トランスケトラーゼはKとして表記し、リブロース−5−リン酸−エピメラーゼはLとして表記し、リボース−5−リン酸エピメラーゼはMとして表記し、キシルキナーゼ(xylukinase)はNとして表記し、キシロースイソメラーゼはOとして表記し、及びキシリトール還元酵素はPとして表記し、リボース−5−リン酸イソメラーゼ(rpi)はQとして表記し、D−リブロース−5−リン酸3−エピメラーゼ(rpe)はRとして表記し、ホスホエノールピルビン酸シンターゼ(pps)はSとして表記し、フルクトースビスリン酸アルドラーゼ(fba)はTとして表記し、EI(ptsI)はUとして表記し、EIICBGlc(ptsG)はVとして表記し、EIIAGlc(crr)はWとして表記し、HPr(ptsH)はXとして表記し、ガラクトース(galP)はYとして表記し、グルコキナーゼ(glk)はZとして表記し、グルコース−6−リン酸脱水素酵素(zwf)はAAとして表記する。上記の通り、ピルビン酸脱炭酸酵素複合体のaceE、aceF、及び/又はlpdA酵素を単独で使用して、又は3つの酵素のうち2つを、又は3つ酵素のうち3つを使用して、ピルビン酸脱炭酸酵素の活性を増加させることができる。したがって、任意の及びすべての酵素の組み合わせは、本明細書においてA−Mとして表記されることが明示的に企図され、並びに任意の及びすべての酵素の組み合わせはA−AAとして表記される。更に、上記の任意の組み合わせを、本明細書に記載の任意の酵素及び/又は酵素経路(例えば、ホスホケトラーゼ、MVA経路ポリペプチド、イソプレンシンターゼ、DXP経路ポリペプチド)と組み合わせて使用することができる。
他の分子的操作を使用して、メバロン酸産生に指向する炭素取り込みを増加させることができる。このような方法のうちの1つは、メバロン酸経路に合流する経路に対する負の制御効果を減少させ、低下させるか、又は除去するものである。例えば、一部の場合では、遺伝子aceEF−lpdAはオペロンであり、pdhRの上流に遺伝子を4つ有する。遺伝子pdhRは、このオペロンの転写に対する負の制御因子である。pdhRは、ピルビン酸の非存在下で標的プロモータに結合し、転写を抑制する。この制御因子は同様の手法でndh及びcyoABCDも制御する(Ogasawara,H.et al.2007.J.Bact.189:5534〜5541)。一態様では、pdhR制御因子を欠失させることにより、ピルビン酸の供給及びそれに伴うメバロン酸、イソプレノイド前駆体、及びイソプレノイドの産生を向上させることができる。
本明細書で使用するとき、用語「最少培地(minimal medium又はminimal media)」は、概して細胞増殖に必要とされる最低限の栄養素を含有している増殖培地を指すが、常に1種以上のアミノ酸(例えば、1、2、3、4、5、6、7、8、9又は10種以上のアミノ酸)が存在していないわけではない。最少培地は、典型的には:(1)細菌を増殖させるための炭素源;(2)細菌種及び増殖条件によって異なる様々な塩;並びに(3)水;を含有する。炭素源は、グルコースなどの単糖から、本明細書で以下により詳細に記載されるような、他のバイオマスのより複雑な加水分解物、例えば、酵母エキスなどといった多様なものであり得る。塩は、概してマグネシウム、窒素、リン及びイオウなどの必須元素を提供し、細胞がタンパク質及び核酸を合成できるようにする。また、最少培地には、特定のプラスミド及び同様物を維持すべく選別するために、抗菌剤などの選択剤を添加することもできる。例えば、微生物が、例えばアンピシリン又はテトラサイクリンなどの特定の抗菌剤に耐性である場合、耐性を欠く細胞の増殖を阻害する目的で培地に抗菌剤を添加することができる。培地には、所望される生理学的又は生化学的特性について選別するのに必要とされる、例えば特定のアミノ酸などといった他の化合物を添加することができる。
本発明の組換え細胞を維持し及び増殖させるのに好適な材料及び方法は、以下、例えば、実施例の節に記載される。微生物培養物の維持及び増殖に好適な他の材料及び方法は当該技術分野において周知である。例示的な手法としては、国際公開第2009/076676号、米国特許出願第第2009/0203102号、国際公開第2010/003007号、米国特許出願第2010/0048964号、国際公開第2009/132220号、米国特許出願第2010/0003716号、Gerhardt et al.,編の一般細菌学に関係する手法についてのマニュアル、American Society for Microbiology,Washington,D.C.(1994)又はBrock in Biotechnology:テキスト「工業微生物学(Industrial Microbiology)」第2版(1989)Sinauer Associates,Inc.(Sunderland,MA)が挙げられる。一部の態様では、細胞は、宿主細胞に挿入された核酸によりコードされている、ホスホケトラーゼポリペプチド、並びに限定するものではないが、mvaE及びmvaS遺伝子産物、イソプレンシンターゼ、DXP経路(例えば、DXS)、IDI、又はPGLポリペプチドなどの、上流及びMVA経路下流由来のその他の酵素の発現を可能にする条件下で培養培地中で培養される。
一部の態様では、本明細書に記載の任意の方法は更に、産生された化合物を回収する工程を包含する。一部の態様では、本明細書に記載の任意の方法は更に、イソプレンを回収する工程を包含する。一部の態様では、イソプレンは吸着ストリッピングにより回収される(例えば、米国特許第2011/0178261号を参照されたい)。一部の態様では、本明細書に記載の任意の方法は更に異種ポリペプチドを回収する工程を包含する。一部の態様では、本明細書に記載の任意の方法は更に、テルペノイド又はカロテノイドを回収する工程を含む。
ビフィドバクテリウム・インファンティス(Bifidobacterium infantis)の染色体DNAをATCCから得た(ATCC# 15697D−5,ATCC(Manassas,VA))。プライマーCMP283:5’−ctgtatTCATGAcgagtcctgttattggcacc−3’(列番号30)及びCMP284:5’−ctctatGAATTCTCACTCGTTGTCGCCAGCG−3’(配列番号31)、テンプレートとして100ngのDNA、並びに製造元に従ってポリメラーゼHerculase II Fusion(Agilent,Santa Clara,CA)を使用して、ホスホケトラーゼ(PKL)酵素をコードしている遺伝子を増幅させた。精製後、消化によりBspHI及びEcoRIにより2798bpの断片を切り出し、NcoI/EcoRIにより消化したpTrcHis2B(Invitrogen,Carlsbad,CA)と連結して、プラスミドpCMP1090を生成した(配列番号15−図6)。
ラクトバチルス・ロイテリ(Lactobacillus reuteri)株F275の染色体DNAをATCC から得た(ATCC# 23272D−5,ATCC(Manassas,VA))。プライマーCMP34:5’−taaggaggaataaacATGGCAGTAGATTACGATTCCAAG−3’(配列番号32)及びCMP335:5’−ttctagaaagcttcgttacttaagacccttccaagtccag−3’(配列番号33)、テンプレートとして100ngのDNA、並びに製造元に従ってポリメラーゼHerculase II Fusion(Agilent(Santa Clara,CA))を使用して、ホスホケトラーゼ(PKL)酵素をコードしている遺伝子を増幅させた。精製後、2442bpの断片を、NcoI/EcoRIにより消化したpTrcHis2B(Invitrogen(Carlsbad,CA))にGENEARTシームレス・クローニング・キット(Invitrogen(Carlsbad,CA))を使用して組み込み、プラスミドpCMP1029を生成した(配列番号16−図7)。
大腸菌(E. coli)株BL21(Novagen)中のクエン酸シンターゼ遺伝子(gltA)の前に位置するプロモータを、常時低発現型プロモータ、すなわちGI1.2(米国特許第7,371,558号)によりあらかじめ置き換えた。2つの野生型プロモータはgltAに関し記載されている(Wilde,R,and J.Guest.1986.J.Gen.Microbiol.132:3239〜3251)。末端プロモータの−35領域に直後に合成プロモータを挿入した。プライマーUpgltACm−F(5’−TATTTAATTTTTAATCATCTAATTTGACAATCATTCAACAAAGTTGTTACAATTAACCCTCACTAAAGGGCGG−3’(配列番号34))及びDngltA1.xgiCm−R(5’−TCAACAGCTGTATCCCCGTTGAGGGTGAGTTTTGCTTTTGTATCAGCCATATATTCCACCAGCTATTTGTTAGTGAATAAAAGTGGTTGAATTATTTGCTCAGGATGTGGCATHGTCAAGGGCTAATACGACTCACTATAGGGCTCG−3’(配列番号35))、及びテンプレートとしてGene Bridges(Heidelberg,Germany)プラスミドFRT−gb2−Cm−FRTを使用し、PCR産物を得た。PCR産物を精製し、λredを用いる組み換えに製造元(Gene Bridges,Heidelberg,Germany)による記載の通りに使用した。更なる評価にあたって、複数種のコロニーを選別した。プロモータ領域は、プライマーgltAPromSeqF:5’−GGCAGTATAGGCTGTTCACAAAATC−3’(配列番号36)及びgltApromSeqR:5’−CTTGACCCAGCGTGCCTTTCAGC−3’(配列番号37)と、テンプレートとしてコロニーから抽出したDNA(コロニーを30μLのH2Oに再懸濁し、95℃で4分加熱し、スピンダウンしたもの。50μLのPCR反応には、この溶液のうち2μLをテンプレートとして使用する)とを使用しPCR増幅させた。得られたPCR産物の配列決定結果を観察した後、GI1.2プロモータ(米国特許第7,371,558号)を保有しているコロニーを、CMP141と命名した。
プラスミドpTrcHis2B、pCMP1090(ビフィドバクテリウム・インファンティス(Bifidobacterium infantis)由来のPKL)及びpCMP1029(ラクトバチルス・ロイテリ(Lactobacillus reuteri)由来のPKL)の存在下で、株CMP674に電気穿孔法を行った。LB+カルベニシリン50μg/mLで、コロニーを単離した各形質転換からコロニーを1つ選択し、それぞれCMP1036、CMP1038及びCMP1040と命名した。
(i)材料
K2HPO4を13.6g、KH2PO4を13.6g、MgSO4 *7H2Oを2g、クエン酸一水和物を2g、クエン酸鉄アンモニウムを0.3g、(NH4)2SO4を3.2g、酵母エキスを0.2g、1000X微量金属溶液を1mL。すべての成分を共に加え、脱イオン水に溶解させる。水酸化アンモニウム(30%)によりpHを6.8に調整し、及び容量を調整する。0.22μmフィルタで培地を濾過滅菌する。pHを調整及び滅菌後にグルコース10.0g及び抗菌剤を加えた。
クエン酸*H2O 40g、MnSO4 *H2O 30g、NaCl 10g、FeSO4*7H2O 1g、CoCl2*6H2O 1g、ZnSO4*7H2O 1g、CuSO4*5H2O 100mg、H3BO3 100mg、NaMoO4*2H2O 100mg。各成分を1成分ずつ脱イオン水に溶解する。pHをHCl/NaOHで3.0に調整した後、溶液を用量に調整し、0.22μmフィルタでフィルタ滅菌する。
ルリア・ベルターニブロス+抗生物質で細胞を一晩生育させた。翌日、OD600が0.05になるよう、50ug/mLのカルベニシリンを含有させた20mLのTM3培地(250mLのバッフル付き三角フラスコ)により細胞を希釈し、34℃かつ200rpmでインキュベートする。2時間生育させた後、OD600を測定し、200uMのIPTGを加える。3.5時間以上経過後、1.5mLのサンプルを遠心分離し、上清を廃棄し、ペレットを100μLの無水冷メタノールに再懸濁した。
アセチル−リン酸を抽出するため、OD 0.57〜2.26へと生育させた大腸菌(E. coli)細胞1.5mLを遠心分離によりスピンダウンし、100μLの無水冷メタノールをペレットに加えた。メタノールにより急冷したサンプルを−20℃で数日間保存した。以降のサンプル加工には、細胞を穏やかに再懸濁し、−9℃で5分間遠心分離し、上清を清潔なバイアル瓶に吸引する工程を包含した。2%酢酸を含有させた75μLの水により、ペレットを2回再抽出した。各抽出後、−9℃にて遠心分離することにより細胞片をペレット化し、全3つの抽出物由来の上清を一緒に集め、1μLのトリブチルアミンを添加した。Thermo Finnigan TSQシステム(Thermo Electron Corporation(San Jose,CA))を使用し、LCMSによるアセチルリン酸の質量分析を実施した。システムの調節、データ収集、及び質量スペクトルデータの評価は、ソフトウェアXCalibur及びLCQuan(Thermo Electron Corp)を使用して行った。移動相を勾配させて、流速0.4mL/分にてSynergi MAX−RP 5μM HPLCカラム(150×2mm,Phenomenex)に加えた。勾配プロファイルは、溶媒Aは15mMトリブチルアミン/10mM酢酸水溶液、溶媒Bはメタノール、及び溶媒Cは水として、99% A及び1% Bをt=0〜1分;80% A及び20% Bをt=11分;75% B及び25% Cをt=12〜14分;99% A及び1% Bをt=15〜16分のように適用した。エレクトロスプレーイオン化質量分析(ESI−MS/MS)ネガティブモード(ESIスプレー電位2.5〜3.0kV、イオントランスファーチューブ温度390℃)でを用い、アセチルリン酸の質量検出を実施したところ、前駆体イオンのm/z値は138.9であった。PO3 −プロダクトイオン(m/z=79.0,衝突エネルギー20V,衝突ガス圧0.22kPa(1.7mTorr),Rt=13.2分)により生成されたピークの積分強度をもとに、アセチルリン酸濃度を求めた。標準アセチルリン酸(Sigma−Aldrich)を注入することにより得られた検量線を使用して、細胞抽出物中の代謝産物濃度を算出した。OD=200の培養液1mLにおける消費に基づき、アセチルリン酸の細胞内濃度を求めた。すべての細胞の総体積は50Mlである(図8)。
ホスホケトラーゼを発現している株では、ホスホケトラーゼを発現しない対照株よりも高濃度のアセチルリン酸細胞内濃度を有した(CMP1040及びCMP1038)(図8)。
プラスミドpMCM82(発現ベクターMCM82(米国特許出願公開番号第2010/0196977号)とともに、プラスミドpTrcHis2B(Invitrogen(Carlsbad,CA))、pCMP1090(ビフィドバクテリウム・インファンティス(Bifidobacterium infantis)由来のPKL)又はpCMP1029(ラクトバチルス・ロイテリ(Lactobacillus reuteri)PKL)を使用して、CMP1047を形質転換させた。宿主CMP1047を対数増殖期の中期まで34℃にてLBで生育させ、電気穿孔に備え、培養物の当量の氷冷ddH2Oにより2回洗浄し、培養物の1/10当量の氷冷ddH2Oに再懸濁した。100μLの細胞懸濁液を1μLの各プラスミドDNAと組み合わせて、2mmの電気穿孔法キュベットに入れ、25uFD、200オーム、2.5kVで電気穿孔し、直ちに1mLのLBにより冷却した。細胞を回収し、34℃にて1時間振盪し、次に50μg/mLのスペクチノマイシン+50μg/mLのカルベニシリンを添加したLBプレートにより34℃にて一晩形質転換体させた。各形質転換用にコロニーを1つ選択し、それぞれCMP1053、1055及び1057と命名した。
(i)材料
K2HPO4を13.6g、KH2PO4を13.6g、MgSO4 *7H2Oを2g、クエン酸一水和物を2g、クエン酸鉄アンモニウムを0.3g、(NH4)2SO4を3.2g、酵母エキスを0.2g、1000X微量金属溶液を1mL。すべての成分を共に加え、脱イオン水に溶解させる。水酸化アンモニウム(30%)によりpHを6.8に調整し、及び容量を調整する。0.22μmフィルタで培地を濾過滅菌する。pH調整及び滅菌後にグルコース10.0g及び抗菌剤を添加する。
クエン酸*H2Oを40g、MnSO4 *H2Oを30g、NaClを10g、FeSO4*7H2Oを1g、CoCl2*6H2Oを1g、ZnSO4*7H2Oを1g、CuSO4*5H2Oを100mg、H3BO3を100mg、NaMoO4*2H2Oを100mg。各成分を1成分ずつ脱イオン水に溶解する。pHをHCl/NaOHで3.0に調整した後、溶液を用量に調整し、0.22μmフィルタでフィルタ滅菌する。
ルリア・ベルターニブロス+抗生物質で細胞を一晩生育させる。翌日、OD600が0.05になるよう、250mLのバッフル付き三角フラスコにおいて細胞を20mLのTM3培地(50μg/mLのスペクチノマイシン及び50μg/mLのカルベニシリンを含有)により希釈し、34℃かつ200rpmでインキュベートする。2時間生育させた後、OD600を測定し、200uMのIPTGを加える。発酵工程の間、規則的に試料を採取する。各時点でOD600を測定する。24時間後、メバロン酸をHPLCにより解析した。次の様式でHPLC解析を実施した:300μLのブロスに54μLの10%(w/v)H2SO4を加え、混合物を氷上で5分インキュベートした。次に、試料を14,000×gで5分遠心分離し、HPLC解析のため上清を回収し、次の条件でHPLC解析を実施した:(1)BioRad−AminexHPX−87Hイオン排除カラム(300mm×7.8mm)(カタログ番号125−0140)(BioRad,Hercules,California);(2)カラム温度=50℃;(3)BioRad−マイクロガードCarbo−H詰め替えカートリッジ(30mm×4.6mm)(カタログ番号125−0129)(BioRad);(4)移動相=0.01N H2SO4;(5)流速=0.6mL/分;(6)およその圧力=約6550.0kPa(950psi);(7)装填量=100μL;(8)実施時間=26分。
ホスホケトラーゼを発現している株は、対照株よりも生育が遅かった(図9)。CMP1057(ラクトバチルス・ロイテリ(L. reuteri)ホスホケトラーゼ遺伝子を発現している)は、対照の空プラスミド又はビフィドバクテリウム・インファンティス(B. infantis)由来のホスホケトラーゼを含有している株と比較して、多量にメバロン酸を産生した(図10)。
MCM521可溶化液を使用し、CMP674にメバロン酸経路の下流を形質導入する(表2を参照されたい)。製造元に従いカナマイシンマーカーを除去する(Gene Bridges、Heidelberg、Germany)。MCM521の下流の経路は、別の遺伝子を使用することにより各遺伝子の前に位置するrbsを改変し、オペロン上流のプロモータを変更することで改変することができる。lacI、イソプレンシンターゼ及びメタノサルシナ・マゼイ(M. mazei)メバロン酸キナーゼを発現している発現プラスミドの、プラスミドpMCM82(発現ベクターMCM82(米国特許出願公開番号第2010/0196977号))及びプラスミドpCMP1090(ビフィドバクテリウム・インファンティス(Bifidobacterium infantis)由来のPKL)、pCMP1029(ラクトバチルス・ロイテリ(Lactobacillus reuteri)由来のPKL)又はpTrcHis2BをCMP1047に電気穿孔した(2段階で)。LB+スペクチノマイシン50μg/mL+カルベニシリン50μg/mL+クロラムフェニコール25μg/mLでコロニーを選別する。
(i)材料
K2HPO4を13.6g、KH2PO4を13.6g、MgSO4 *7H2Oを2g、クエン酸一水和物を2g、クエン酸鉄アンモニウムを0.3g、(NH4)2SO4を3.2g、酵母エキスを0.2g、1000X微量金属溶液を1mL。すべての成分を共に加え、脱イオン水に溶解させる。水酸化アンモニウム(30%)によりpHを6.8に調整し、及び容量を調整する。0.22μmフィルタで培地を濾過滅菌する。pH調整及び滅菌後にグルコース10.0g及び抗菌剤を添加する。
クエン酸*H2Oを40g、MnSO4 *H2Oを30g、NaClを10g、FeSO4*7H2Oを1g、CoCl2*6H2Oを1g、ZnSO4*7H2Oを1g、CuSO4 *5H2Oを100mg、H3BO3を100mg、NaMoO4 *2H2Oを100mg。各成分を1成分ずつ脱イオン水に溶解する。pHをHCl/NaOHで3.0に調整した後、溶液を用量に調整し、0.22μmフィルタでフィルタ滅菌する。
ルリア・ベルターニブロス+抗生物質で細胞を一晩生育させる。翌日、OD600が0.1になるよう、250mLのバッフル付き三角フラスコにおいて細胞を20mLのTM3培地(50μg/mLのスペクチノマイシン、25μg/mLのクロラムフェニコール、及び50μg/mLカルベニシリンを含有)により希釈し、34℃かつ200rpmでインキュベートする。2時間生育させた後、OD600を測定し、200uMのIPTGを加える。発酵工程の間、規則的に試料を採取する。各時点でOD600を測定する。また、ガスクロマトグラフ−マススペクトロメータ(GC−MS)(Agilent)によるヘッドスペース解析によりイソプレンのオフガス分析を行う。100μLの全ブロスをGCバイアルに入れ密閉し、34℃かつ200rpmで30分間インキュベートする。70℃で7分のインキュベーションからなる熱殺菌工程後、試料をGCに装填する。記録される比産生量は、イソプレン量のGCによる読み取り値(μg/L)を、インキュベート時間(30分)及びOD600測定値で除算した値である。
ホスホケトラーゼを発現している株は、ホスホケトラーゼを発現しない対照株よりも生育が遅かった。ホスホケトラーゼポリペプチドを発現している株において、イソプレンの比産生性、収率、CPI、及び/又は力価の増加が観察されたことから、ホスホケトラーゼポリペプチドを発現している株は、対照の空プラスミド(すなわち、ホスホケトラーゼを発現しない株)を含有している株と比較して、イソプレンの産生の増強を示す。
MCM521可溶化液を使用し、CMP674にメバロン酸経路の下流を形質導入する(表2を参照されたい)。製造元に従いカナマイシンマーカーを除去する(Gene Bridges、Heidelberg、Germany)。MCM521の下流の経路は、別の遺伝子を使用することにより各遺伝子の前に位置するrbsを改変し、オペロン上流のプロモータを変更することで改変することができる。染色体上のプロモータ及び/又はrbsを変更させるか、又はプラスミドから発現させるかのいずれかにより、ファルネシル二リン酸シンターゼ(ispA)を過剰発現させる。
(i)材料
K2HPO4を13.6g、KH2PO4を13.6g、MgSO4 *7H2Oを2g、クエン酸一水和物を2g、クエン酸鉄アンモニウムを0.3g、(NH4)2SO4を3.2g、酵母エキスを0.2g、1000X微量金属溶液を1mL。すべての成分を共に加え、脱イオン水に溶解させる。水酸化アンモニウム(30%)によりpHを6.8に調整し、及び容量を調整する。次に、0.22μmのフィルタを用い培地を濾過滅菌した。滅菌及びpH調整後にグルコース10.0g及び抗菌剤を添加する。
クエン酸*H2Oを40g、MnSO4 *H2Oを30g、NaClを10g、FeSO4*7H2Oを1g、CoCl2*6H2Oを1g、ZnSO4*7H2Oを1g、CuSO4 *5H2Oを100mg、H3BO3を100mg、NaMoO4 *2H2Oを100mg。各成分を1成分ずつ脱イオン水に溶解する。pHをHCl/NaOHで3.0に調整した後、溶液を用量に調整し、0.22μmフィルタでフィルタ滅菌する。
ルリア・ベルターニブロス+抗生物質で細胞を一晩生育させる。翌日、OD600が0.05になるよう、250mLのバッフル付き三角フラスコにおいて細胞を20mLのTM3培地(50μg/mLのスペクチノマイシン、25μg/mLのクロラムフェニコール、及び50μg/mLカルベニシリンを含有)により希釈し、34℃かつ200rpmでインキュベートする。接種前に、各培養フラスコには20%(v/v)ドデカン(Sigma−Aldrich)を積層し、これまでに記載のとおり、揮発性セスキテルペン産物を捕捉する(Newman et.al.,2006)。
ホスホケトラーゼポリペプチドを発現している株を、同様のバックグラウンドを持ち、空プラスミド(すなわち、ホスホケトラーゼポリペプチドを不含有である)を含有している株と比較する。ホスホケトラーゼポリペプチドを発現している株において、アモルファジエン又はファルネセンの比産生性、収率、CPI、及び/又は力価の増加が観察されたことから、ホスホケトラーゼポリペプチドを発現している株は、対照の空プラスミド(すなわち、ホスホケトラーゼを発現しない株)を含有している株と比較して、アモルファジエン又はファルネセンの産生の増強を示す。
Newman,J.D.,Marshal,J.L.,Chang,M.C.Y.,Nowroozi,F.,Paradise,E.M.,Pitera,D.J.,Newman,K.L.,Keasling,J.D.,2006.High−level production of amorpha−4,11−diene in a two−phase partitioning bioreactor of metabolically engineered E.coli.Biotechnol.Bioeng.95,684〜691。
ホスホケトラーゼポリペプチドをコードしている異種遺伝子並びにメバロン酸経路由来の遺伝子を発現している大腸菌(E. coli)においてメバロン酸産生を評価し、15L規模で流加式培養により生育させた。
培地組成(発酵培地1Lあたり):
K2HPO4(7.5g)、MgSO4*7H2O(2g)、クエン酸一水和物(2g)、クエン酸鉄アンモニウム(0.3g)、酵母エキス(0.5g)、50%硫酸(1.6mL)、1000X改変微量金属溶液(1mL)。すべての成分を共に加え、脱イオン水に溶解させた。この溶液を加熱滅菌した(123℃で20分)。水酸化アンモニウム(28%)によりpHを7.0に調整し、容量にメスアップした。滅菌及びpH調製後にグルコース10g、ビタミン溶液8mL、及び抗生物質を加えた。
クエン酸*H2O(40g)、MnSO4*H2O(30g)、NaCl(10g)、FeSO4*7H2O(1g)、CoCl2*6H2O(1g)、ZnSO*7H2O(1g)、CuSO4*5H2O(100mg)、H3BO3(100mg)、NaMoO4*2H2O(100mg)。各成分を1つずつ脱イオン水に溶解させ、HCl/NaOHによりpHを3.0に調整し、次に溶液を用量に調整し、0.22μmのフィルタを用いろ過滅菌した。
チアミン塩酸塩(1.0g)、D−(+)−ビオチン(1.0g)、ニコチン酸(1.0g)、塩酸ピリドキシン(4.0g)。各成分を1つずつ脱イオン水に溶解させ、HCl/NaOHによりpHを3.0に調整し、次に溶液を用量に調整し、孔径0.22μmのフィルタを用いろ過滅菌した。
グルコース(0.590kg)、脱イオン水(0.393kg)、K2HPO4(7.4g)及び100% Foamblast882(8.9g)。すべての成分を合わせて混合し、オートクレーブ処理した。
MgSO4*7H2O(296g)、クエン酸水和物(296g)、クエン酸鉄アンモニウム(49.6g)。すべての成分を水に溶解させ、用量に調整し、0.22μmのフィルタを用い、ろ過滅菌した。更なる添加物は加えずに、このうち16.8mLを培養槽に直接加えた後に滅菌した。
4時間間隔でブロス試料を回収し、発酵ブロス中のメバロン酸濃度をHPLC分析により測定した。屈折計の測定値と、予め作成した検量線とを比較して、ブロス試料中のメバロン酸濃度を測定した。
システム:Waters Alliance 2695
カラム:BioRad−AminexHPX−87Hイオン排除カラム300mm×7.8mm(カタログ番号125−0140
カラム温度:50℃
ガードカラム:詰め替え用BioRad−Microguard Cation H(30mM×4.6mM)カタログ番号125−0129
ランニング緩衝液:0.01NのH2SO4;
ランニング緩衝液流速:0.6mL/分
最適流圧:約7.5〜8.2MPa(約1100〜1200psi)
注入容量:20μL
検出器:屈折率(Knauer K−2301)
実行時間:26分
ホスホケトラーゼを発現している株(CMP1057)により発酵させた場合、グルコースに対するメバロン酸収率は、空プラスミド対照株(CMP1053)よりも高かった。下記図11〜13及び表3を参照されたい。加えて、ホスホケトラーゼを発現している株(CMP1057)を有する発酵ブロスでは、空プラスミドの対照株(CMP1053)と比較して、発酵ブロス中の酢酸の蓄積は少なかった。図14を参照されたい。
Life Technologiesの酵母発現系(コンピテントな野生型サッカロミセス・セレビシエ(S. cerevisiae)(INVSc1(カタログ番号C810−00))及びプラスミドpYES2/CT(カタログ番号V8251−20)を使用して、イソプレンシンターゼ、MVA経路上流、MVA経路下流及びホスホケトラーゼを発現させるための上記の各種遺伝子コンストラクトを調製する。
ATCC15697株、ビフィドバクテリウム・ロングム(Bifidobacterium longum)亜種のインファンティス(infantis)の染色体DNAをATCC(Manassas,VA)から得た。プライマーCMP283:5’−ctgtatTCATGAcgagtcctgttattggcacc−3’(配列番号42)及びCMP284:5’−ctctatGAATTCTCACTCGTTGTCGCCAGCG−3’(配列番号43)、並びに製造元のプロトコルに従ってポリメラーゼHerculase(Life Technologies(Carlsbad,CA))を使用して、ビフィドバクテリウム・ロンガム(B. longhum)PKL(配列番号3)をコードしている遺伝子をポリメラーゼ連鎖反応(PCR)により染色体DNAから増幅させた。EcoRI及びBspHIによりPCR産物を消化した後、精製した。精製後、約2500bpの断片を、EcoRI/NcoIにより消化したpTrcHis2B(Invitrogen(Carlsbad,CA))にGENEARTシームレス・クローニング・キット(Invitrogen(Carlsbad,CA))を使用して組み込み、プラスミドpCMP1090を生成した(配列番号15−図6)。
CMP1133株(BL21,Δpgl PL.2mKKDyl,GI1.2gltA,yhfSFRTPyddVIspAyhfS,thiFRTtruncIspA)を形質転換してPKL発現株を構築し、20μg/mLのカナマイシンを含有させたルリア・ベルターニプレートでコロニーを選別した。製造元により推奨されるプロトコルを用い、カナマイシンマーカーを除去して(Gene Bridges,Heidelberg,Germany)記載の株を作製した(表4)。
TrcPKLビフィドバクテリウム・ロングム(B. longum)(CMP1183株)、pTrcPKLエンテロコッカス・ガリナラム(E. gallinarum)(CMP1328株)、pTrcPKLクロストリジウム・アセトブチリカム(C. acetobutylicum)(CMP1328株)、pTrcPKLラクトバチルス・ロイテリ(L. reuteri)(CMP1182株)、pTrcPKLのストック・パンクチホルム(N. punctiforme)(CMP1308株)、pTrcPKLロードシュードモナス・パルストリス(R. palustris)(CMP1309)、pTrcPKLパントエア(Pantaoea)(CMP1331)、pTrcPKLムチラギニバクター・パルジス(M. paludis)(CMP1330)、及びpTrcPKLサーモビフィダ・フスカ(T. fusca)(CMP1333)発現株をLB培地で生育し、OD600が約0.5の時点で200μMのIPTGにより発現誘導し、並びに30℃又は34℃の温度で4時間発現誘導した。4mLの培養ブロスを3000rpmにて10分間遠心分離して細胞を回収した。細胞ペレットを、0.1% DNAase及び0.5mMのAEBSFを添加した2mLの50mM MES/50mM NaCl(pH 6.0)に再懸濁した。96.5MPa(14,000psi)にて、フレンチ・プレス・セル(American Instrument Company)により細胞懸濁液を溶解した。次に、Eppendorf 5804R遠心機により、可溶化液を4℃にて15,000RPMで10分遠心分離した。上清及びペレットを分離した。ペレットを溶解液(50mM MES/50mM NaCl(pH 6.0)緩衝液)に再懸濁した。上清及びペレットサンプルを4〜12% SDS−PAGEゲル電気泳動により分析した。ホスホケトラーゼの可溶性画分対ペレット(不溶性)画分を比較することにより溶解度を評価した。
以降の動態試験において使用するため、ビフィドバクテリウム・ロングム(B. longum)由来のホスホケトラーゼ(PKL)を精製した。ビフィドバクテリウム・ロングム(B. longum)由来のPKLを、pTrc His2Bプラスミドを入れたBL21株(CMP1183)において発現させた。細胞を、50μg/mLカルベニシリンを添加したルリア・ベルターニ培地にて34℃にて生育させた後、誘導を行った。200μM IPTGによる誘導後、培地を室温の振盪器に移し、5時間置いた。4℃、10,000rpmにて10分間遠心分離して細胞を回収した。精製前に−80℃にて細胞ペレットを保存した。精製に際し、ビフィドバクテリウム・ロングム(B. longum)PKL細胞ペレットを20mM HEPES(pH 7.0)、60mM NaCl、0.5mM AEBSF、0.5mM MgCl2、0.1mg/mL DNaseIに再懸濁した。フレンチ・プレス・セルを繰り返し通過させて細胞を溶解し、50,000rpmで30分間、超遠心により清澄化させた。ビフィドバクテリウム・ロングム(B. longum)由来のPKLを含有している清澄化させた溶解液を、まずは、50mM Tris/50mM NaCl(pH 7)により平衡化させたMonoQ 10/100GLカラム(GE Healthcare)に装填し、50mM Tris/1 M NaCl(pH 7)までび勾配を用い溶出した。得られた画分をSDS−PAGEにより分析した。ビフィドバクテリウム・ロングム(B. longum)PKLを、50mM Tris/50mM NaCl(pH 7)により平衡化したSuperdex 200 10/300GL及びMonoQ 10/300GL(pH 6.0)を使用し、50mM MES/50mM NaCl〜1M NaClの勾配を用い、更に精製した。A280を用いビフィドバクテリウム・ロングム(B. longum)PKLを定量し、149550(ベクターNTIにより測定した)のモル吸光係数を求め、及びまたゲルデンシトメトリー法によっても求めた。イオン交換及びゲル濾過クロマトグラフィーを用い精製し、見かけの均一性>95%でPKLを調製した。ヒドロキサム酸第二鉄による分析を用いPKL活性を解析するのに使用するため、ビフィドバクテリウム・ロングム(B. longum)PKLを含有している画分をプールした。
フルクトース6−リン酸(陽性対照)、リボース5−リン酸(陰性対照)、又はセドヘプツロース7−リン酸を単独で、又はビフィドバクテリウム・ロングム(B. longum)ホスホケトラーゼとともに発現している細胞を生育させ、LC−MS検出により、代謝産物の産生について解析した。
チオラーゼ、HMG−CoAシンターゼ、及びHMG−CoA還元酵素(表7)をコードする遺伝子に加え、ビフィドバクテリウム・ロンガム(Bifidobacterium longum)、エンテロコッカス・ガリナラム(Enterococcus gallinarum)、クロストリジウム・アセトブチリカム(Clostridium acetubutylicum)、ノストック(Nostoc)、ロードシュードモナス・パルストリス(Rhodopseudomonas palustris)、パントエア(Pantoea)、又はサーモビフィダ・フスカ(Thermobifida fusca)由来のホスホケトラーゼを発現させることにより、メバロン酸(MVA)を産生している大腸菌(E. coli)株を作製した。ホスホケトラーゼを発現しないMVA産生株を対照として使用した(表7)。小規模実験において、グルコース存在下で生育させた場合のホスホケトラーゼ発現及びメバロン酸収率を、ホスホケトラーゼを発現しない対照株と比較して、ホスホケトラーゼ発現株を選別した。
酵母エキス及びMgSO4を不含有の改変TM3培地の組成(発酵培地1Lあたり):
K2HPO4を13.6g、KH2PO4を13.6g、クエン酸一水和物を2g、クエン酸鉄アンモニウムを0.3g、(NH4)2SO4を3.2g、1000X微量金属溶液を1mL。すべての成分を共に加え、脱イオン水に溶解させる。水酸化アンモニウム(30%)によりpHを6.8に調整し、及び容量を調整する。0.22μmフィルタで培地を濾過滅菌する。pHを調整及び滅菌後にグルコース10.0g及び抗菌剤を加えた。
クエン酸*H2O(40g)、MnSO4*H2O(30g)、NaCl(10g)、FeSO4*7H2O(1g)、CoCl2*6H2O(1g)、ZnSO*7H2O(1g)、CuSO4*5H2O(100mg)、H3BO3(100mg)、NaMoO4*2H2O(100mg)。各成分を1つずつ脱イオン水に溶解させ、HCl/NaOHによりpHを3.0に調整し、次に溶液を用量に調整し、0.22μmのフィルタを用いろ過滅菌した。
増殖速度の測定
必要に応じて抗生物質を添加したLB培地を3mL入れた振盪チューブに、培養物のグリセロールストックを接種した。培養物を、30℃にて約15時間、220rpmでインキュベートした。TM3培地(MgSO4及び酵母エキスを不含有)、1%グルコース、8mM MgSO4、0.02%酵母エキス及び必要に応じて抗生物質を組み合わせて、追加のTM3培地を調製した。最終的なOD600が0.2になるよう、48ウェル滅菌ブロックの各ウェルに追加のTM3を2mL加えた。ブロックをBreathe Easierメンブレンによりシールし、34℃にて2時間、600rpmでインキュベートした。2時間生育させた後、マイクロタイタープレートにおいてOD600を測定し、各種濃度のIPTGにより細胞に発現誘導した。IPTGによる発現誘導から4時間にわたって、1時間毎にOD600の読み取りを行った。OD600の測定は、SpectraMax Plus190(Molecular Devices)を使用して行った。細胞を一晩生育させ、OD600を測定した。
300μLの細胞培養物を96ウェルコニカルプレートにおいて4℃にて3000rpmで10分間遠心分離し、グルコースサンプルを回収した。上清を脱イオン水で10倍希釈し、Pointe Scientificから購入したグルコースオキシダーゼ解析キットを用いグルコース濃度測定した。
34μLの10%硫酸と300μLの細胞培養物とを組み合わせ、氷上で10分間インキュベートし、メバロン酸サンプルを加工した。4℃にて10分後、混合物を4℃、3000rpmにて10分間遠心分離した。250μLの上清を96ウェルコニカルプレートに回収し、HPLCによるメバロン酸測定のため、Zone−Free(商標)Filmsプレートシーラーによりシールした。グルコース消費量に対し総メバロン酸量を算出することによりメバロン酸収率を求めた。
発現解析のため、発現誘導から4時間の時点の細胞培養全ブロスのうち50μLのサンプルを、50μLの2X SDSサンプル緩衝液とともに95℃で5分間ボイルし、10μLのサンプルを4〜12% Bis−Trisゲルに装填した。精製したホスホケトラーゼ酵素及び前染色した標準を各ゲルに加えた。GelをSimplyBlue Coomassie(登録商標)G−250染色により染色し、脱イオン水で脱染色した。
4mLの培養ブロスを3000rpmにて10分間遠心分離して細胞を回収した。細胞ペレットを、0.1% DNAase及び0.5mM AEBSFを添加した2mLの100mM Tris、100mM NaCl(pH 7.6)に再懸濁した。96.5MPa(14,000psi)にて、フレンチ・プレス・セル(American Instrument Company)により細胞懸濁液を溶解した。次に、Eppendorf 5804R遠心機により、可溶化液を4℃にて15,000RPMで10分遠心分離した。上清及びペレットを分離した。ペレットを溶解緩衝液に再懸濁した。ペレット及び上清の両方を調製するためにゲルサンプルを調製し、電気泳動を実施した。iBlot(登録商標)ドライ・ブロッティング・システムを使用し、タンパク質をニトロセルロース膜に転写した後、1:10,000希釈したウサギ抗ビフィドバクテリウム・ロンガム(Bifido longum)ホスホケトラーゼ血清を一次抗体とし、2μg/mL濃度のAlexa Fluor 488ヤギ抗ウサギIgGを検出抗体として、製造元のプロトコルに従って免疫検出した。Storm 860分子イメージャ(Molecular Dynamics)を使用して、青色蛍光スキャナスクリーンを用い、タンパク質のバンドを検出し、ソフトウェアImageQuantを使用してタンパク質濃度を算出した。
ビフィドバクテリウム・ロングム(B. longum)のPKL(EWL1319)、エンテロコッカス・ガリナラム(E. gallinarum)のPKL(EWL1341)、ノストック(Nostoc)のPKL(EWL1344)、ロードシュードモナス・パルストリス(R. palustris)PKL(EWL1347)、パントエア(Pantoea)PKL(EWL1350)、又はサーモビフィダ・フスカ(T. fusca)PKL(EWL1353)を発現するよう遺伝子操作された大腸菌(E. coli)株によるMVA産生の解析により、発現誘導に使用するIPTGの増加と、メバロン酸収率の増加とが相関することが実証された(図46)。PKLを発現するよう遺伝子操作された株から調製した全細胞溶解液におけるタンパク質発現の解析により、IPTGによりタンパク質の発現が誘導されたことが示された(図47A〜D)。これらの所見により、メバロン酸収率の増加は、ホスホケトラーゼタンパク質の発現が増加した結果であることが示される。PKLを発現しないMVA産生対照株(CHL875)と比較した場合のメバロン酸収率の増加は、ビフィドバクテリウム・ロングム(B. longum)PKL(EWL1319)、エンテロコッカス・ガリナラム(E. gallinarum)PKL(EWL1341)、又はクロストリジウム・アセトブチリカム(C. acetobutylicum)PKL(EWL1359)発現株においても観察された。ビフィドバクテリウム・ロングム(B. longum)PKL(図48A)、ガリナラム(gallinarum)PKL(図49A)、又はクロストリジウム・アセトブチリカム(C. acetobutylicum)PKL(図50A)発現株におけるメバロン酸収率の増加は、IPTGの発現誘導濃度の増加と相関した。ビフィドバクテリウム・ロングム(B. longum)PKL、エンテロコッカス・ガリナラム(E. gallinarum)PKL、及びクロストリジウム・アセトブチリカム(C. acetobutylicum)のホスホケトラーゼ発現株のメバロン酸収率は、対照株CHL875により産生される最大メバロン酸収率と比較して増加していた。PKLを発現している遺伝子操作株から調製した細胞溶解液におけるタンパク質解析により、ビフィドバクテリウム・ロングム(B. longum)PKL(図48B及びC)、ガリナラム(gallinarum)PKL(図49B)、又はクロストリジウム・アセトブチリカム(C. acetobutylicum)のPKL(図50B)は、IPTGによる発現誘導により発現されることが確認された。ビフィドバクテリウム・ロングム(B. longum)のPKL発現株から単離された上清画分及びペレット画分の更なる解析により、ホスホケトラーゼは、不溶性画分(図48C)と比較して、主に可溶性画分(図48B)に存在することが示された。これらの結果は、メバロン酸収率の増加は、ホスホケトラーゼ発現が増加したことによる結果だという結論と一致する。
エンテロコッカス・ガリナラム(Enterococcus gallinarum)(EWL1341株)及びクロストリジウム・アセトブチリカム(Clostridium acetubutylicum)(EWL1359株)由来のホスホケトラーゼを発現しているメバロン酸(MVA)産生株を、ホスホケトラーゼを発現しないMVA産生株(CHL875株)と、15規模の実験において比較した。グルコースに対する累積MVA収率、グルコースに対する瞬間収率、MVAの体積産生性、MVAの比産生性及び細胞性能指数(CPI)を測定し、解析した。
培地組成(発酵培地1Lあたり):
K2HPO4(7.5g)、MgSO4*7H2O(2g)、クエン酸一水和物(2g)、クエン酸鉄アンモニウム(0.3g)、酵母エキス(0.5g)、50%硫酸(1.6mL)、1000X改変微量金属溶液(1mL)。すべての成分を共に加え、脱イオン水に溶解させた。この溶液を加熱滅菌した(123℃で20分)。水酸化アンモニウム(28%)によりpHを7.0に調整し、容量にメスアップした。滅菌及びpH調製後にグルコース10g、ビタミン溶液8mL、及び抗生物質を加えた。
クエン酸*H2O(40g)、MnSO4*H2O(30g)、NaCl(10g)、FeSO4*7H2O(1g)、CoCl2*6H2O(1g)、ZnSO*7H2O(1g)、CuSO4*5H2O(100mg)、H3BO3(100mg)、NaMoO4*2H2O(100mg)。各成分を1つずつ脱イオン水に溶解させ、HCl/NaOHによりpHを3.0に調整し、次に溶液を用量に調整し、0.22μmのフィルタを用いろ過滅菌した。
チアミン塩酸塩(1.0g)、D−(+)−ビオチン(1.0g)、ニコチン酸(1.0g)、塩酸ピリドキシン(4.0g)。各成分を1つずつ脱イオン水に溶解させ、HCl/NaOHによりpHを3.0に調整し、次に溶液を用量に調整し、孔径0.22μmのフィルタを用いろ過滅菌した。
MgSO4*7H2O(296g)、クエン酸水和物(296g)、クエン酸鉄アンモニウム(49.6g)。すべての成分を水に溶解させ、用量に調整し、0.22μmのフィルタを用い、ろ過滅菌した。
グルコース(0.590kg)、脱イオン水(0.393kg)、K2HPO4(7.4g)及び100% Foamblast882(8.9g)。すべての成分を合わせて混合し、オートクレーブ処理した。供給溶液をオートクレーブにより処理した後、滅菌フード中で、供給ボトルに栄養塩類を添加する。滅菌後に供給溶液に添加するものは(供給溶液1kg当たり)マクロ塩溶液5.54mL、ビタミン溶液6.55mL、1000X改変微量金属溶液0.82mLである。
導入されたメバロン酸経路由来の遺伝子及びエンテロコッカス・ガリナラム(E. gllinarum)(EWL1341株)又はクロストリジウム・アセトブチリカム(C. acetobutylicum)(EWL1359株)のいずれかから単離されたPKLを発現している改変大腸菌(E. coli)(BL21)宿主(CMP1133)によるメバロン酸(MVA)産生を、この株を15L規模の流加式培養において生育させることにより評価した(表9)。導入されたメバロン酸経路由来の遺伝子を発現しているもののPKL(CHL875株)は発現しない改変大腸菌(E. coli)(BL21)宿主(CMP1133)と比較して、MVA産生を比較し、ホスホケトラーゼの使用により収率が増加するかを決定した。
グルコースに対する収率(重量%)=総MVA(t)/[(供給重量(0)−供給重量(t)+83.5)*0.59)],
式中、0.59グルコース供給溶液中のグルコースの割合(重量%)であり、及び83.5は、t=0の時点で発酵槽に供給したバッチのg重量である。各供給量は、独立して重量%として測定した。
CPI=総MVA量(g)/総乾燥細胞重量
システム:Waters Alliance 2695
カラム:BioRad−Aminex HPX−87Hイオン排除カラム300mm×7.8mm(カタログ番号125−0140
カラム温度:50℃
ガードカラム:詰め替え用BioRad−Microguard Cation H(30mm×4.6mm)カタログ番号125−0129
ランニング緩衝液:0.01NのH2SO4;
ランニング緩衝液流速:0.6mL/分
最適流圧:約7.5〜8.2MPa(約1100〜1200psi)
注入容量:20μL検出器:屈折率(Knauer K−2301)
実行時間:26分
クロストリジウム・アセトブチリカム(C. acetobutylicum)ホスホケトラーゼの発現株(EWL1359、実施番号20121056、201201057、20121058)は、ホスホケトラーゼ非発現株よりも(CHL875、実施番号20120821、20120976、20121059)グルコースに対するMVAの累積収率(%)が高かった(表11及び図51)。少なくとも100μMのIPTGを槽に加えた場合に(20121057及び20121058)、MVA収率の増加が顕著であった。
ビフィドバクテリウム・ロンガム(Bifidobacterium longum)、エンテロコッカス・ガリナラム(Enterococcus gallinarum)、又はクロストリジウム・アセトブチリカム(Clostridium acetobutylicum)由来のホスホケトラーゼを発現させて、イソプレン産生大腸菌(E. coli)株を作製した。ホスホケトラーゼを発現していないイソプレン産生株を対照として使用した(表13)。小規模実験において、グルコース存在下で生育させた場合のホスホケトラーゼ発現及びイソプレン収率を、ホスホケトラーゼを発現しない対照株と比較して、ホスホケトラーゼ発現株を選別した。
TM3培地組成(発酵培地1L当たり):
K2HPO4を13.6g、KH2PO4を13.6g、MgSO4 *7H2Oを2g、クエン酸一水和物を2g、クエン酸鉄アンモニウムを0.3g、(NH4)2SO4を3.2g、酵母エキスを0.2g、1000X微量金属溶液を1mL。すべての成分を共に加え、脱イオン水に溶解させる。水酸化アンモニウム(30%)によりpHを6.8に調整し、及び容量を調整する。0.22μmフィルタで培地を濾過滅菌する。pHを調整及び滅菌後にグルコース10.0g及び抗菌剤を加えた。
K2HPO4を13.6g、KH2PO4を13.6g、クエン酸一水和物を2g、クエン酸鉄アンモニウムを0.3g、(NH4)2SO4を3.2g、1000X微量金属溶液を1mL。すべての成分を共に加え、脱イオン水に溶解させる。水酸化アンモニウム(30%)によりpHを6.8に調整し、及び容量を調整する。0.22μmフィルタで培地を濾過滅菌する。pHを調整及び滅菌後にグルコース10.0g及び抗菌剤を加えた。
クエン酸*H2O(40g)、MnSO4*H2O(30g)、NaCl(10g)、FeSO4*7H2O(1g)、CoCl2*6H2O(1g)、ZnSO*7H2O(1g)、CuSO4*5H2O(100mg)、H3BO3(100mg)、NaMoO4*2H2O(100mg)。各成分を1つずつ脱イオン水に溶解させ、HCl/NaOHによりpHを3.0に調整し、次に溶液を用量に調整し、0.22μmのフィルタを用いろ過滅菌した。
増殖速度の測定
必要に応じて抗生物質を添加したLB培地を3mL入れた振盪チューブに、培養物のグリセロールストックを接種した。培養物を、30℃にて約15時間、220rpmでインキュベートした。TM3培地(MgSO4及び酵母エキスを不含有)、1%グルコース、8mM MgSO4、0.02%酵母エキス及び必要に応じて抗生物質を組み合わせて、追加のTM3培地を調製した。最終的なOD600が0.2になるよう、48ウェル滅菌ブロックの各ウェルに追加のTM3を2mL加えた。ブロックをBreathe Easierメンブレンによりシールし、34℃にて2時間、600rpmでインキュベートした。2時間生育させた後、マイクロタイタープレートにおいてOD600を測定し、各種濃度のIPTGにより細胞に発現誘導した。IPTGによる発現誘導から4時間にわたって、1時間毎にOD600の読み取りを行った。OD600の測定は、SpectraMax Plus190(Molecular Devices)を使用して行った。細胞を一晩生育させ、OD600を測定した。
300μLの細胞培養物を96ウェルコニカルプレートにおいて4℃にて3000rpmで10分間遠心分離し、グルコースサンプルを回収した。上清を脱イオン水で10倍希釈し、Pointe Scientificから購入したグルコースオキシダーゼ解析キットを用いグルコース濃度測定した。
IPTGによる発現誘導後、4時間にわたって1時間毎に100μLのイソプレンサンプルを96ウェルガラスブロックに回収した。アルミホイルによりガラスブロックをシールし、Thermomixerを使用し、34℃にて、450rpmで振盪しながら30分間インキュベートした。30分後、ブロックを70℃の水浴に2時間保持し、ガスクロマトグラフィー質量分析法を使用し、ヘッドスペース測定法において、イソプレン濃度を決定した。
発現解析のため、発現誘導から4時間の時点の細胞培養全ブロスのうち50μLのサンプルを、50μLの2X SDSサンプル緩衝液とともに95℃で5分間ボイルし、10μLのサンプルを4〜12% Bis−Trisゲルに装填した。精製したホスホケトラーゼ酵素及び前染色した標準を各ゲルに加えた。GelをSimplyBlue Coomassie(登録商標)G−250染色により染色し、脱イオン水で脱染色した。
メタノコッカシド・バートニイ(M. Burtonii)のメバロン酸キナーゼの発現存在下でビフィドバクテリウム・ロングム(B. longum)PKLを(EWL1427株)、メタノコッカシド・バートニイ(M. Burtonii)メバロン酸キナーゼの発現存在下でエンテロコッカス・ガリナラム(E. gallinarum)PKLを(EWL1430株)、又はメタノコッカシド・バートニイ(M. Burtonii)メバロン酸キナーゼの発現存在下でクロストリジウム・アセトブチリカム(C. acetobutylicum)PKLを(EWL1446株)、発現するよう遺伝子操作された大腸菌(E. coli)株により、グルコースから産生されたイソプレンを、PKLを発現しない対照株(MCM2158株)と比較して分析したところ、発現誘導に使用するIPTGの増加と、イソプレン収率の増加とが相関することが実証された(図62A及びB)。PKLを発現するよう遺伝子操作された株から調製した全細胞溶解液におけるタンパク質発現の解析により、IPTGによりタンパク質の発現が誘導されたことが示された(図63A及びB)。これらの所見により、イソプレン収率の増加は、ホスホケトラーゼタンパク質の発現が増加した結果であることが示される。
ビフィドバクテリウム・ロングム(B. longum)(EWL1427株)又はエンテロコッカス・ガリナラム(E. gallinarum)(EWL1430株)由来のホスホケトラーゼを発現するイソプレン産生株を、ホスホケトラーゼを発現しないイソプレン産生株(MCM2158株)と、15L規模の実験において、イソプレン産生について比較した。グルコースに対する累積イソプレン収率、グルコースに対するイソプレン瞬間収率及び細胞性能指数(CPI)を測定し、解析した。
培地組成(発酵培地1Lあたり):
K2HPO4(7.5g)、MgSO4*7H2O(2g)、クエン酸一水和物(2g)、クエン酸鉄アンモニウム(0.3g)、酵母エキス(0.5g)、50%硫酸(1.6mL)、1000X改変微量金属溶液(1mL)。すべての成分を共に加え、脱イオン水に溶解させた。この溶液を加熱滅菌した(123℃で20分)。水酸化アンモニウム(28%)によりpHを7.0に調整し、容量にメスアップした。滅菌及びpH調製後にグルコース10g、ビタミン溶液8mL、及び抗生物質を加えた。
クエン酸*H2O(40g)、MnSO4*H2O(30g)、NaCl(10g)、FeSO4*7H2O(1g)、CoCl2*6H2O(1g)、ZnSO*7H2O(1g)、CuSO4*5H2O(100mg)、H3BO3(100mg)、NaMoO4*2H2O(100mg)。各成分を1つずつ脱イオン水に溶解させ、HCl/NaOHによりpHを3.0に調整し、次に溶液を用量に調整し、0.22μmのフィルタを用いろ過滅菌した。
チアミン塩酸塩(1.0g)、D−(+)−ビオチン(1.0g)、ニコチン酸(1.0g)、塩酸ピリドキシン(4.0g)。各成分を1つずつ脱イオン水に溶解させ、HCl/NaOHによりpHを3.0に調整し、次に溶液を用量に調整し、孔径0.22μmのフィルタを用いろ過滅菌した。
MgSO4*7H2O(296g)、クエン酸水和物(296g)、クエン酸鉄アンモニウム(49.6g)。すべての成分を水に溶解させ、用量に調整し、0.22μmのフィルタを用い、ろ過滅菌した。
グルコース(0.590kg)、脱イオン水(0.393kg)、K2HPO4(7.4g)及び100% Foamblast882(8.9g)。すべての成分を合わせて混合し、オートクレーブ処理した。供給溶液をオートクレーブにより処理した後、滅菌フード中で、供給ボトルに栄養塩類を添加する。滅菌後に供給溶液に添加するものは(供給溶液1kg当たり)マクロ塩溶液5.54mL、ビタミン溶液6.55mL、1000X改変微量金属溶液0.82mLである。
メバロン酸経路由来の導入遺伝子及びビフィドバクテリウム・ロングム(B. longum)(EWL1427株)又はエンテロコッカス・ガリナラム(E. gallinarum)(EWL1430株)のいずれかから単離されたPKLを発現している改変大腸菌(E. coli)(BL21)宿主(MCM2065)によるイソプレン生産を、株を15L規模の流加式培養において生育させることにより評価した(表14)。導入されたメバロン酸経路由来の遺伝子を発現しているもののPKL(MCM2158株)は発現しない改変大腸菌(E. coli)(BL21)宿主(MCM2065)と、イソプレン産生を比較し、ホスホケトラーゼの使用により収率が増加するかを決定した。
グルコースに対する収率(重量%)=総イソプレン(t)/[(供給重量(0)−供給重量(t)+83.5)*0.59)],
式中、0.59グルコース供給溶液中のグルコースの割合(重量%)であり、及び83.5は、t=0の時点で発酵槽に供給したバッチのg重量である。各供給量は、独立して重量%として測定した。
イソプレン瞬間収率(g/g%)=産生されたイソプレン(t1−t0)/消費されたグルコース(t1−t0)*100
CPI=総イソプレン量(g)/総乾燥細胞重量
システム:Waters Alliance 2695
カラム:BioRad−AminexHPX−87Hイオン排除カラム300mm×7.8mm(カタログ番号125−0140
カラム温度:50℃
ガードカラム:詰め替え用BioRad−Microguard Cation H(30mm×4.6mm)カタログ番号125−0129
ランニング緩衝液:0.01NのH2SO4;
ランニング緩衝液流速:0.6mL/分
最適流圧:約7.5〜8.2MPa(約1100〜1200psi)
注入容量:20μL
検出器:屈折率(Knauer K−2301)
実行時間:26分
エンテロコッカス・ガリナラム(E. gallinarum)ホスホケトラーゼ(EWL1340、20121136)の発現株は、ホスホケトラーゼ非発現株よりも(MCM2158、20121134)グルコースに対するイソプレンの累積収率(%)が高かった(表16及び図65)。エンテロコッカス・ガリナラム(E. gallinarum)ホスホケトラーゼ(EWL1430、20121136)発現株は、ホスホケトラーゼ非発現株(MCM2158、20121134)よりもイソプレン瞬間収率(%)が高く(表16及び図66)、及びより長期間にわたって高い累積収率を維持したことから累積収率が高くなった(表16及び図65)。ビフィドバクテリウム・ロンガム(B. longum)ホスホケトラーゼ発現株(EWL1427、20121135)は、誘導実験においてより高い瞬間収率を有していたものの(図66)、この株は最高収率に達するまでにより長時間を要し、かつ終了時のグルコースに対するイソプレンの累積収率は、対照株(MCM2158、20121134)のものとおおよそ同一であった(表16及び図65)。
エンテロコッカス・ガリナラム(E. gallinarum)(表19及び表21)、クロストリジウム・アセトブチリカム(C. acetobutylicum)(表20)、又はビフィドバクテリウム・ロンガム(B. longum)(表21)由来のホスホケトラーゼ(PKL)発現株を動態試験により解析した。試料を調製する際、細胞ペレットは、14Lの培養工程中に1mL培養物から得て−80℃で保管した。1mg/mLのリゾチーム、0.2mg/mLのDNaseI、及び0.5mMのAEBSFを添加した50mM MES(pH 6)にペレット試料を再懸濁し、OD(600)=20に揃えた。ODを揃えた細胞ペレットを、培養液の割合に対し4.8MPa(700psi)に設定したフレンチプレスMini Cellに繰り返し通過させて溶解した。次に、溶解した試料を、4℃にて14,000rpmで10分間遠心分離して清澄化させた。溶解物の可溶性画分の活性解析及びタンパク質測定を実施した。0.5〜0.05mg/mLの範囲の濃度でのBSA滴定をもとに作成した標準曲線を使用し、Bradford法(バイオラッド)により総タンパク質を測定した。
上記の通りに活性ホスホケトラーゼポリペプチドを含むイソプレン産生株を更に遺伝子操作して、次の遺伝子:リボース−5−リン酸イソメラーゼ(rpiA及び/又はrpiB)、D−リブロース−5−リン酸3−エピメラーゼ(rpe)、トランスケトラーゼ(tktA及び/又はtktB)、トランスアルドラーゼB(tal B)、ホスホエノールピルビン酸シンターゼ(ppsA)、リン酸アセチルトランスフェラーゼ(pta及び/又はeutD)のうちの1つ以上の活性を向上させて、ホスホケトラーゼ経路に取り込まれる炭素量を増加させることができる(図72)。特定の態様では、次の遺伝子、rpiA、rpiB、rpe、tktA、tktB、talB、ppsA、eutD、及び/又はptaの活性は、染色体上のプロモータ及び/又はrbsを変更することにより増加させることができ、又はプラスミドに発現させることにより増加させることができる。一実施形態では、リボース−5−リン酸イソメラーゼ(rpiA及び/又はrpiB)の活性は、染色体上のプロモータ及び/又はrbsを変更することにより増加させることができ、又はプラスミドに発現させることにより増加させることができる。他の実施形態では、D−リブロース−5−リン酸3−エピメラーゼ(rpe)の活性は、染色体上のプロモータ及び/又はrbsを変更することにより増加させることができ、又はプラスミドに発現させることにより増加させることができる。他の実施形態では、トランスケトラーゼ(tktA及び/又はtktB)の活性は、染色体上のプロモータ及び/又はrbsを変更することにより増加させることができ、又はプラスミドに発現させることにより増加させることができる。更に別の実施形態では、トランスアルドラーゼB(tal B)の活性は、染色体上のプロモータ及び/又はrbsを変更することにより増加させることができ、又はプラスミドに発現させることにより増加させることができる。他の実施形態では、ホスホエノールピルビン酸シンターゼ(ppsA)の活性は、染色体上のプロモータ及び/又はrbsを変更することにより増加させることができ、又はプラスミドに発現させることにより増加させることができる。更に他の実施形態では、リン酸アセチルトランスフェラーゼ(pta及び/又はeutD)の活性は、染色体上のプロモータ及び/又はrbsを変更することにより増加させることができ、又はプラスミドに発現させることにより増加させることができる。特定の態様では、次の遺伝子、rpiA、rpiB、rpe、tktA、tktB、tal B、ppsA、eutD、及び/又はptaのイソ酵素は、染色体上のプロモータ及び/又はrbsを変更することにより増加させることができ、又はプラスミドに発現させることにより増加させることができる。
(i)材料
K2HPO4を13.6g、KH2PO4を13.6g、MgSO4 *7H2Oを2g、クエン酸一水和物を2g、クエン酸鉄アンモニウムを0.3g、(NH4)2SO4を3.2g、酵母エキスを0.2g、1000×微量金属溶液を1mL。すべての成分を共に加え、脱イオン水に溶解させる。水酸化アンモニウム(30%)によりpHを6.8に調整し、及び容量を調整する。0.22μmフィルタで培地を濾過滅菌する。pH調整及び滅菌後にグルコース10.0g及び抗菌剤を添加する。
クエン酸*H2O 40g、MnSO4 *H2O 30g、NaCl 10g、FeSO4 *7H2O 1g、CoCl2 *6H2O 1g、ZnSO4 *7H2O 1g、CuSO4 *5H2O 100mg、H3BO3 100mg、NaMoO4 *2H2O 100mg。各成分を1成分ずつ脱イオン水に溶解する。pHをHCl/NaOHで3.0に調整した後、溶液を用量に調整し、0.22μmフィルタでフィルタ滅菌する。
ルリア・ベルターニブロス+抗生物質で細胞を一晩生育させる。翌日、OD600が0.1になるよう、250mLのバッフル付き三角フラスコにおいて細胞を20mLのTM3培地(50μg/mLのスペクチノマイシン、25μg/mLのクロラムフェニコール、及び50μg/mLカルベニシリンを含有)により希釈し、34℃かつ200rpmでインキュベートする。2時間生育させた後、OD600を測定し、200uMのIPTGを加える。発酵工程の間、規則的に試料を採取する。各時点でOD600を測定する。また、ガスクロマトグラフ−マススペクトロメータ(GC−MS)(Agilent)によるヘッドスペース解析によりイソプレンのオフガス分析を行う。100μLの全ブロスをGCバイアルに入れ密閉し、34℃かつ200rpmで30分間インキュベートする。70℃で7分のインキュベーションからなる熱殺菌工程後、試料をGCに装填する。記録される比産生量は、イソプレン量のGCによる読み取り値(μg/L)を、インキュベート時間(30分)及びOD600測定値で除算した値である。
(i)材料
K2HPO4(7.5g)、MgSO4*7H2O(2g)、クエン酸一水和物(2g)、クエン酸鉄アンモニウム(0.3g)、酵母エキス(0.5g)、50%硫酸(1.6mL)、1000X改変微量金属溶液(1mL)。すべての成分を共に加え、脱イオン水に溶解させた。この溶液を加熱滅菌した(123℃で20分)。水酸化アンモニウム(28%)によりpHを7.0に調整し、容量にメスアップした。滅菌及びpH調製後にグルコース10g、ビタミン溶液8mL、及び抗生物質を加えた。
クエン酸*H2O(40g)、MnSO4*H2O(30g)、NaCl(10g)、FeSO4*7H2O(1g)、CoCl2*6H2O(1g)、ZnSO*7H2O(1g)、CuSO4*5H2O(100mg)、H3BO3(100mg)、NaMoO4*2H2O(100mg)。各成分を1つずつ脱イオン水に溶解させ、HCl/NaOHによりpHを3.0に調整し、次に溶液を用量に調整し、0.22μmのフィルタを用いろ過滅菌した。
チアミン塩酸塩(1.0g)、D−(+)−ビオチン(1.0g)、ニコチン酸(1.0g)、塩酸ピリドキシン(4.0g)。各成分を1つずつ脱イオン水に溶解させ、HCl/NaOHによりpHを3.0に調整し、次に溶液を用量に調整し、孔径0.22μmのフィルタを用いろ過滅菌した。
MgSO4*7H2O(296g)、クエン酸水和物(296g)、クエン酸鉄アンモニウム(49.6g)。すべての成分を水に溶解させ、用量に調整し、0.22μmのフィルタを用い、ろ過滅菌した。
グルコース(0.590kg)、脱イオン水(0.393kg)、K2HPO4(7.4g)及び100% Foamblast882(8.9g)。すべての成分を合わせて混合し、オートクレーブ処理した。供給溶液をオートクレーブにより処理した後、滅菌フード中で、供給ボトルに栄養塩類を添加する。滅菌後に供給溶液に添加するものは(供給溶液1kg当たり)マクロ塩溶液5.54mL、ビタミン溶液6.55mL、1000X改変微量金属溶液0.82mLである。
2種の質量分析器iSCAN(ハミルトン・サンドストランド)、及びHiden HPR20(Hiden Analytical)により、排出気体中の、イソプレン、酸素、窒素、及び二酸化炭素濃度を別個に測定した。
Claims (28)
- イソプレンを産生することのできる組換え細菌または酵母細胞であって、
前記組換え細胞は、ラクトバチルス、ビフィドバクテリウム、エンテロコッカスまたはクロストリジウム由来のホスホケトラーゼ活性を有するポリペプチドをコードしている1種以上の異種核酸と、(i)完全なMVA経路の1種以上のポリペプチドをコードしている1種以上の核酸、及び(ii)イソプレンシンターゼポリペプチドをコードしている異種核酸と、を含み、
好適な培地で前記組換え細胞を培養することにより、イソプレンの産生が得られ、及び
前記組換え細胞は、ホスホケトラーゼ活性を有するポリペプチドをコードしている1種以上の異種核酸を含まないイソプレン産生細胞と比較して多量のイソプレンを産生する、
組換え細胞。 - イソプレノイド前駆体を産生することのできる組換え細菌または酵母細胞であって、
前記組換え細胞は、ラクトバチルス、ビフィドバクテリウム、エンテロコッカスまたはクロストリジウム由来のホスホケトラーゼ活性を有するポリペプチドをコードしている1種以上の異種核酸と、完全なMVA経路の1種以上のポリペプチドをコードしている1種以上の異種核酸とを含み、
好適な培地で前記組換え細胞を培養することによりイソプレノイド前駆体の産生が得られ、
前記組換え細胞は、ホスホケトラーゼ活性を有するポリペプチドをコードしている1種以上の異種核酸を含まないイソプレノイド前駆体産生細胞と比較して多量のイソプレノイド前駆体を産生し、及び
前記イソプレノイド前駆体は、メバロン酸、イソペンテニルピロリン酸(IPP)、又はジメチルアリル二リン酸(DMAPP)の1種以上である、
組換え細胞。 - イソプレノイドを産生することのできる組換え細菌または酵母細胞であって、
前記組換え細胞は、ラクトバチルス、ビフィドバクテリウム、エンテロコッカスまたはクロストリジウム由来のホスホケトラーゼ活性を有するポリペプチドをコードしている1種以上の異種核酸と、(i)完全なMVA経路の1種以上のポリペプチドをコードしている1種以上の核酸及び(ii)ポリプレニルピロリン酸シンターゼポリペプチドをコードしている異種核酸とを含み、
好適な培地で前記組換え細胞を培養することにより、イソプレノイドの産生が得られ、及び
前記組換え細胞は、ホスホケトラーゼ活性を有するポリペプチドをコードしている1種以上の異種核酸を含まないイソプレノイド産生細胞と比較して多量のイソプレノイドを産生する、
組換え細胞。 - ホスホケトラーゼ活性を有するポリペプチドが、キシルロース5−リン酸からグリセルアルデヒド3−リン酸及びアセチルリン酸を合成することができる、請求項1〜3のいずれか一項に記載の細胞。
- ホスホケトラーゼ活性を有するポリペプチドが、フルクトース6−リン酸からエリスロース4−リン酸及びアセチルリン酸を合成することができる、請求項1〜3のいずれか一項に記載の細胞。
- ホスホケトラーゼ活性を有するポリペプチドが、ビフィドバクテリウム・ロングム(Bifidobacterium longum)、エンテロコッカス・ガリニアラム(Enterococcus galliniarum)、クロストリジウム・アセトブチリカム(Clostridium acetobutilicum)、ビフィドバクテリウム・ブレビ(Bifidobacterium breve)、ビフィドバクテリウム・アニマリス(Bifidobacterium animalis)、ラクトバチルス・プランタラム(Lactobacillus plantarum)、又はラクトバチルス・ロイテリ(Lactobacillus reuteri)由来である、請求項1〜3のいずれか一項に記載の細胞。
- ホスホケトラーゼ活性を有するポリペプチドが、ビフィドバクテリウム・ロングム(Bifidobacterium longum)、エンテロコッカス・ガリニアラム(Enterococcus galliniarum)、又はクロストリジウム・アセトブチリカム(Clostridium acetobutilicum)由来である、請求項1〜3のいずれか一項に記載の細胞。
- イソプレンシンターゼポリペプチドが、植物のイソプレンシンターゼポリペプチドである、請求項1に記載の細胞。
- 前記イソプレンシンターゼポリペプチドが、クズ属(Pueraria)又はハコヤナギ属(Populus)、又はウラジロハコヤナギとヤマナラシの交雑種(Populus alba x Populus tremula)由来のポリペプチドである、請求項1に記載の細胞。
- 前記イソプレンシンターゼポリペプチドが、プエラリア・モンタナ(Pueraria montana)又はプエラリア・ロバタ(Pueraria lobata)、ヤマナラシ(Populus tremuloides)、ウラジロハコヤナギ(Populus alba)、ポプラ・ニグラ(Populus nigra)、又はポプラ・トリコカルパ(Populus trichocarpa)由来である、請求項1に記載の細胞。
- 前記完全なMVA経路の1種以上のポリペプチドが、(a)2分子のアセチルCoAを
縮合させてアセトアセチルCoAを生成する酵素;(b)アセトアセチル−CoAをアセチル−CoAと縮合させてHMG−CoAを生成する酵素(例えば、HMGシンターゼ);(c)HMG−CoAをメバロン酸に変換する酵素;(d)メバロン酸をメバロン酸5−リン酸にリン酸化する酵素;(e)メバロン酸5−リン酸を5−ジホスホメバロン酸に変換する酵素;及び(f)5−ジホスホメバロン酸をイソペンテニルピロリン酸に変換する酵素から選択される、請求項1〜3のいずれか一項に記載の細胞。 - 前記組換え細胞が、1−デオキシ−D−キシルロース5−リン酸(DXP)経路の1種以上のポリペプチドをコードしている1種以上の核酸を更に含む、請求項11に記載の細胞。
- 前記1種以上の核酸が、誘導型プロモータ又は構成型プロモータ下に配置される、請求項1〜3のいずれか一項に記載の細胞。
- 前記1種以上の核酸が、1つ以上の多コピープラスミドにクローニングされる、請求項1〜3のいずれか一項に記載の細胞。
- 前記1種以上の核酸が、前記細胞の染色体に組み込まれる、請求項1〜3のいずれか一項に記載の細胞。
- 前記組換え細胞が、グラム陽性菌細胞またはグラム陰性菌細胞である、請求項1〜3のいずれか一項に記載の細胞。
- 前記組換え細胞が、バチルス・スブチリス(Bacillus subtilis)、ストレプトマイセス・リビダンス(Streptomyces lividans)、ストレプトマイセス・セリカラー(Streptomyces coelicolor)、ストレプトマイセス・グリセウス(Streptomyces griseus)、大腸菌(Escherichia coli)、パントエア・シトレア(Pantoea citrea)、サッカロミセス・セレビシエ(Saccharomyces cerevisieae)、及びヤロウイア・リポリティカ(Yarrowia lipolytica)からなる群から選択される、請求項1〜3のいずれか一項に記載の細胞。
- 前記イソプレノイドが、モノテルペン、ジテルペン、トリテルペン、テトラテルペン、セスキテルペン(sequiterpene)及びポリテルペンからなる群から選択される、請求項3に記載の細胞。
- 前記イソプレノイドが、セスキテルペンである、請求項3に記載の細胞。
- 前記イソプレノイドが、アビエタジエン、アモルファジエン、カレン、α−ファルネセン(famesene)、β−ファルネセン、ファルネソール、ゲラニオール、ゲラニルゲラニオール、リナロール、リモネン、ミルセン、ネロリドール、オシメン、パチョロール、β−ピネン、サビネン、γ−テルピネン、テルピンデン(terpindene)及びバレンセンからなる群から選択される、請求項3に記載の細胞。
- 前記組換え細胞が、酢酸キナーゼ遺伝子の発現が減少するように又は酢酸キナーゼ遺伝子を欠失させるように、更に遺伝子操作されている、請求項1〜3のいずれか一項に記載の細胞。
- 前記組換え細胞が、ホスホトランスアセチラーゼ遺伝子の活性が増加するように更に遺伝子操作されている、請求項1〜3のいずれか一項に記載の細胞。
- イソプレンの生産量が、ホスホケトラーゼ活性を有するポリペプチドをコードしている1種以上の異種核酸を含まない細胞と比較して、少なくとも5%又は少なくとも10%増加している、請求項1に記載の細胞。
- イソプレノイド前駆体の生産量が、ホスホケトラーゼ活性を有するポリペプチドをコードしている1種以上の異種核酸を含まない細胞と比較して、少なくとも5%又は少なくとも10%増加している、請求項2に記載の細胞。
- イソプレノイドの生産量が、ホスホケトラーゼ活性を有するポリペプチドをコードしている1種以上の異種核酸を含まない細胞と比較して、少なくとも5%又は少なくとも10%増加している、請求項3に記載の細胞。
- イソプレン産生法であって、(a)イソプレン産生に好適な条件下で請求項1に記載の組換え細胞を培養する工程、及び(b)イソプレンを産生させる工程、を含む、方法。
- イソプレノイド前駆体の産生法であって、(a)イソプレノイド前駆体の産生に好適な条件下で請求項2に記載の組換え細胞を培養する工程、及び(b)イソプレノイド前駆体を産生させる工程、を含む、方法。
- イソプレノイドの産生法であって、(a)イソプレノイドの産生に好適な条件下で請求項3に記載の組換え細胞を培養する工程、及び(b)イソプレノイドを産生させる工程、を含む、方法。
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201161545083P | 2011-10-07 | 2011-10-07 | |
| US61/545,083 | 2011-10-07 | ||
| PCT/US2012/059136 WO2013066568A1 (en) | 2011-10-07 | 2012-10-05 | Utilization of phosphoketolase in the production of mevalonate, isoprenoid precursors, and isoprene |
Related Child Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2018192631A Division JP6639606B2 (ja) | 2011-10-07 | 2018-10-11 | メバロン酸、イソプレノイド前駆体、及びイソプレンの産生におけるホスホケトラーゼの使用 |
Publications (3)
| Publication Number | Publication Date |
|---|---|
| JP2014531904A JP2014531904A (ja) | 2014-12-04 |
| JP2014531904A5 JP2014531904A5 (ja) | 2015-11-26 |
| JP6420147B2 true JP6420147B2 (ja) | 2018-11-07 |
Family
ID=47046886
Family Applications (2)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2014534810A Active JP6420147B2 (ja) | 2011-10-07 | 2012-10-05 | メバロン酸、イソプレノイド前駆体、及びイソプレンの産生におけるホスホケトラーゼの使用 |
| JP2018192631A Active JP6639606B2 (ja) | 2011-10-07 | 2018-10-11 | メバロン酸、イソプレノイド前駆体、及びイソプレンの産生におけるホスホケトラーゼの使用 |
Family Applications After (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2018192631A Active JP6639606B2 (ja) | 2011-10-07 | 2018-10-11 | メバロン酸、イソプレノイド前駆体、及びイソプレンの産生におけるホスホケトラーゼの使用 |
Country Status (12)
| Country | Link |
|---|---|
| US (3) | US8993305B2 (ja) |
| EP (2) | EP3339426B1 (ja) |
| JP (2) | JP6420147B2 (ja) |
| CN (1) | CN104053765A (ja) |
| AU (1) | AU2012332996A1 (ja) |
| BR (1) | BR112014008278A2 (ja) |
| CA (1) | CA2851265A1 (ja) |
| DK (2) | DK2764084T4 (ja) |
| HK (1) | HK1201071A1 (ja) |
| MX (1) | MX2014004209A (ja) |
| SG (1) | SG11201401249PA (ja) |
| WO (1) | WO2013066568A1 (ja) |
Families Citing this family (38)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US8647642B2 (en) | 2008-09-18 | 2014-02-11 | Aviex Technologies, Llc | Live bacterial vaccines resistant to carbon dioxide (CO2), acidic PH and/or osmolarity for viral infection prophylaxis or treatment |
| JP5387756B2 (ja) * | 2011-01-20 | 2014-01-15 | トヨタ自動車株式会社 | 組換え酵母及びこれを用いた物質生産方法 |
| ES2554814T3 (es) * | 2011-07-12 | 2015-12-23 | Scientist Of Fortune S.A. | Microorganismo recombinante para la producción de metabolitos útiles |
| SG11201401249PA (en) * | 2011-10-07 | 2014-05-29 | Danisco Us Inc | Utilization of phosphoketolase in the production of mevalonate, isoprenoid precursors, and isoprene |
| EP2794851A2 (en) * | 2011-12-23 | 2014-10-29 | Danisco US Inc. | Enhanced production of isoprene using marine bacterial cells |
| WO2013181647A2 (en) | 2012-06-01 | 2013-12-05 | Danisco Us Inc. | Compositions and methods of producing isoprene and/or industrrial bio-products using anaerobic microorganisms |
| WO2014100726A2 (en) | 2012-12-21 | 2014-06-26 | Danisco Us Inc. | Production of isoprene, isoprenoid, and isoprenoid precursors using an alternative lower mevalonate pathway |
| US9518278B2 (en) | 2013-03-14 | 2016-12-13 | The Regents Of The University Of California | Recombinant microorganisms having a methanol elongation cycle (MEC) |
| EP3425049A1 (en) * | 2013-04-10 | 2019-01-09 | Danisco US Inc. | Phosphoketolases for improved production of acetyl coenzyme a-derived metabolites, isoprene, isoprenoid precursors, and isoprenoids |
| WO2015002977A1 (en) * | 2013-07-01 | 2015-01-08 | Braskem S/A Ap 09 | Modified microorganisms and methods of using same for anaerobic coproduction of isoprene and acetic acid |
| EP3043879B1 (en) | 2013-09-12 | 2024-07-03 | Gradiant Corporation | Systems including a condensing apparatus such as a bubble column condenser |
| US10808262B2 (en) | 2013-12-03 | 2020-10-20 | Genomatica, Inc. | Microorganisms and methods for improving product yields on methanol using acetyl-CoA synthesis |
| WO2015127305A2 (en) | 2014-02-20 | 2015-08-27 | Danisco Us Inc. | Recombinant microorganisms for the enhanced production of mevalonate, isoprene, isoprenoid precursors, isoprenoids, and acetyl-coa-derived products |
| WO2015191638A1 (en) * | 2014-06-09 | 2015-12-17 | Massachusetts Institute Of Technology | Dynamic knockdown of central metabolism for redirecting glucose-6-phosphate fluxes |
| BR112017013576A2 (pt) * | 2014-12-30 | 2018-03-06 | Novogy Inc | produção melhorada de lipídios de núcleo em leveduras oleaginosas |
| WO2016134381A1 (en) | 2015-02-20 | 2016-08-25 | The Regents Of The University Of California | Novel host cells and methods for producing isopentenol from mevalonate |
| CN107531763B (zh) * | 2016-02-04 | 2020-11-20 | 中国科学院微生物研究所 | 一种用于生物合成异戊二烯和异戊烯的酶及其突变体 |
| CN107815424B (zh) * | 2016-09-12 | 2021-03-05 | 华东理工大学 | 一种产柠檬烯的解脂耶氏酵母基因工程菌及其应用 |
| US11180535B1 (en) | 2016-12-07 | 2021-11-23 | David Gordon Bermudes | Saccharide binding, tumor penetration, and cytotoxic antitumor chimeric peptides from therapeutic bacteria |
| US11129906B1 (en) | 2016-12-07 | 2021-09-28 | David Gordon Bermudes | Chimeric protein toxins for expression by therapeutic bacteria |
| CA3047268A1 (en) * | 2016-12-16 | 2018-06-21 | Danisco Us Inc. | Bifunctional phosphoketolase-phosphotransacetylase fusion polypeptides |
| CN109234216B (zh) * | 2017-07-10 | 2022-05-20 | 上海医药工业研究院 | 一种生产鲨烯的基因工程菌及其方法 |
| CN108179114B (zh) * | 2017-11-27 | 2021-08-20 | 南京晓庄学院 | 产抗厌氧菌化合物的菌株和发酵方法、抗厌氧菌化合物提取及制备方法和使用方法 |
| WO2019113387A1 (en) | 2017-12-07 | 2019-06-13 | Zymergen Inc. | Engineered biosynthetic pathways for production of (6e)-8-hydroxygeraniol by fermentation |
| KR20200111172A (ko) | 2017-12-21 | 2020-09-28 | 지머젠 인코포레이티드 | 네페탈락톨 산화 환원 효소, 네페탈락톨 합성 효소, 및 네페탈락톤을 생산할 수 있는 미생물 |
| CA3090055A1 (en) * | 2018-03-01 | 2019-09-06 | Total Raffinage Chimie | Metabolic pathways with increased carbon yield |
| CN109486720B (zh) * | 2018-12-18 | 2020-08-04 | 江南大学 | 一种产l-木酮糖的枯草芽孢杆菌及其应用 |
| EP3908663A1 (en) * | 2019-02-20 | 2021-11-17 | Braskem S.A. | Microorganisms and methods for the production of oxygenated compounds from hexoses |
| CN113710798A (zh) * | 2019-02-20 | 2021-11-26 | 布拉斯科公司 | 戊糖和己糖的降解途径 |
| CN109929883A (zh) * | 2019-04-02 | 2019-06-25 | 烟台华康荣赞生物科技有限公司 | 重组酵母、构建方法和其在制备酪醇及衍生物中的应用 |
| KR102229379B1 (ko) * | 2019-06-11 | 2021-03-19 | 주식회사 제노포커스 | 파이토플루엔 고생산성 재조합 미생물 및 이를 이용한 파이토플루엔의 제조방법 |
| WO2021007575A1 (en) * | 2019-07-11 | 2021-01-14 | Bp Corporation North America Inc. | Gluconate dehydratase enzymes and recombinant cells |
| JP6917434B2 (ja) * | 2019-10-25 | 2021-08-11 | 長瀬産業株式会社 | 好気性菌の代謝酵素破壊株およびその培養法 |
| CN112300952B (zh) * | 2020-11-27 | 2023-05-09 | 华东理工大学 | 一种产α-蒎烯的解脂耶氏酵母基因工程菌及其应用 |
| WO2022232444A1 (en) * | 2021-04-29 | 2022-11-03 | Ginkgo Bioworks, Inc. | Genetically modified yeast cells and methods of use for increased lipid yield |
| CN113549562B (zh) * | 2021-07-14 | 2023-10-24 | 南京师范大学 | 一种高效生产广藿香醇的工程菌及其构建方法和应用 |
| WO2023028459A1 (en) * | 2021-08-24 | 2023-03-02 | Lanzatech, Inc. | Microbial fermentation for the production of isoprenoid alcohols and derivatives |
| CN115948456B (zh) * | 2022-10-18 | 2024-09-17 | 重庆大学 | 一种可提升广藿香醇合成量的融合基因及方法 |
Family Cites Families (50)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| DE19629568C1 (de) | 1996-07-15 | 1998-01-08 | Fraunhofer Ges Forschung | Verfahren zur Herstellung von Isopren |
| GB9614979D0 (en) | 1996-07-17 | 1996-09-04 | Newteam Ltd | Aerating arrangement primarily for a shower head |
| US6531303B1 (en) * | 1998-07-06 | 2003-03-11 | Arkion Life Sciences Llc | Method of producing geranylgeraniol |
| EP2292776A1 (en) * | 2000-07-31 | 2011-03-09 | Danisco US Inc. | Manipulation of genes of the mevalonate and isoprenoid pathways to create novel traits in transgenic organisms |
| US7172886B2 (en) | 2001-12-06 | 2007-02-06 | The Regents Of The University Of California | Biosynthesis of isopentenyl pyrophosphate |
| SE0200857D0 (sv) * | 2002-03-19 | 2002-03-19 | Forskarpatent I Syd Ab | Metabolic engineering for improved xylose utilisation of saccharomyces cerevisiae |
| WO2003095651A1 (fr) | 2002-05-10 | 2003-11-20 | Kyowa Hakko Kogyo Co., Ltd. | Procede de production d'acide mevalonique |
| JP4592418B2 (ja) | 2002-10-04 | 2010-12-01 | イー・アイ・デュポン・ドウ・ヌムール・アンド・カンパニー | 高収率での1,3−プロパンジオールの生物生産のための方法 |
| JP4168870B2 (ja) | 2003-08-14 | 2008-10-22 | トヨタ自動車株式会社 | プレニルアルコールの製造方法 |
| JP5590769B2 (ja) | 2004-07-27 | 2014-09-17 | ザ リージェンツ オブ ザ ユニバーシティ オブ カリフォルニア | イソプレノイド化合物を生産するための遺伝的に修飾された宿主細胞および同宿主細胞の使用方法 |
| RU2004124226A (ru) * | 2004-08-10 | 2006-01-27 | Закрытое акционерное общество "Научно-исследовательский институт Аджиномото-Генетика" (ЗАО АГРИ) (RU) | Использование фосфокетолазы для продукции полезных метаболитов |
| BRPI0514201B1 (pt) * | 2004-08-10 | 2020-09-15 | Ajinomoto Co., Inc | Bactéria, e, método para produzir um metabólito útil |
| WO2007024718A2 (en) † | 2005-08-19 | 2007-03-01 | The Regents Of The University Of California | Genetically modified host cells and use of same for producing isoprenoid compounds |
| EP3031924A1 (en) | 2006-05-26 | 2016-06-15 | Amyris, Inc. | Production of isoprenoids |
| US7947478B2 (en) | 2006-06-29 | 2011-05-24 | The Regents Of The University Of California | Short chain volatile hydrocarbon production using genetically engineered microalgae, cyanobacteria or bacteria |
| JP4986547B2 (ja) | 2006-09-04 | 2012-07-25 | 株式会社Adeka | 新規なアセトアセチルCoA合成酵素、それをコードするDNA配列、当該酵素の製造方法および当該酵素を利用したメバロン酸の製造方法 |
| WO2009005704A1 (en) * | 2007-07-03 | 2009-01-08 | The Regents Of The University Of California | Methods of increasing isoprenoid or isoprenoid precursor production |
| BRPI0816951B1 (pt) * | 2007-09-20 | 2023-01-17 | Amyris, Inc | Método para produzir um composto isoprenóide |
| EP2062967A1 (en) * | 2007-11-21 | 2009-05-27 | Technical University of Denmark | Genetically engineered aspergillus |
| DK2235190T3 (en) | 2007-12-13 | 2018-07-23 | Danisco Us Inc | COMPOSITIONS AND PROCEDURES FOR PRODUCING ISOPRENE |
| WO2009100231A2 (en) | 2008-02-06 | 2009-08-13 | The Regents Of The University Of California | Short chain volatile isoprene hydrocarbon production using the mevalonic acid pathway in genetically engineered yeast and fungi |
| EP2706111A1 (en) * | 2008-03-03 | 2014-03-12 | Joule Unlimited Technologies, Inc. | Engineered CO2 fixing microorganisms producing carbon-based products of interest |
| US8173410B2 (en) | 2008-04-23 | 2012-05-08 | Danisco Us Inc. | Isoprene synthase variants for improved microbial production of isoprene |
| US7632995B2 (en) | 2008-05-09 | 2009-12-15 | Monsanto Technology Llc | Plants and seeds of hybrid corn variety CH852179 |
| ES2716231T3 (es) | 2008-07-02 | 2019-06-11 | Danisco Us Inc | Procedimientos para producir isopreno |
| HU3516U (en) | 2008-07-31 | 2008-11-28 | Zoltan Kuthi | Anti-theft protection of draining for fuel tanks |
| MY156588A (en) | 2008-09-15 | 2016-03-15 | Danisco Us Inc | Systems using cell culture for production of isoprene |
| WO2010031068A1 (en) | 2008-09-15 | 2010-03-18 | Danisco Us Inc. | Reduction of carbon dioxide emission during isoprene production by fermentation |
| SG169614A1 (en) † | 2008-09-15 | 2011-04-29 | Goodyear Tire & Rubber | Conversion of prenyl derivatives to isoprene |
| MY169163A (en) | 2008-09-15 | 2019-02-19 | Danisco Us Inc | Increased isoprene production using the archaeal lower mevalonate pathway |
| KR20110122672A (ko) | 2008-12-30 | 2011-11-10 | 다니스코 유에스 인크. | 이소프렌 및 공-산물을 제조하는 방법 |
| US20100297749A1 (en) | 2009-04-21 | 2010-11-25 | Sapphire Energy, Inc. | Methods and systems for biofuel production |
| US8518686B2 (en) | 2009-04-23 | 2013-08-27 | Danisco Us Inc. | Three-dimensional structure of isoprene synthase and its use thereof for generating variants |
| JP4760951B2 (ja) | 2009-05-08 | 2011-08-31 | トヨタ自動車株式会社 | ブタノール生産能を有する組換え微生物及びブタノールの製造方法 |
| TWI427149B (zh) * | 2009-06-17 | 2014-02-21 | Danisco Us Inc | 使用dxp及mva途徑之改良之異戊二烯製造 |
| TWI434921B (zh) | 2009-06-17 | 2014-04-21 | Danisco Us Inc | 從生物異戊二烯組合物製造燃料成分之方法及系統 |
| US20120252054A1 (en) | 2009-09-15 | 2012-10-04 | Sapphire Energy Inc | System for transformation of the chloroplast genome of scenedesmus sp. and dunaliella sp. |
| BR112012006939A2 (pt) † | 2009-09-29 | 2015-09-08 | Butamax Tm Advanced Biofuels | célula hospedeira de produção de levedura recombinante e método para a produção de um produto selecionado do grupo que consiste em 2,3-butanodiol, isobutanol, 2-butanol, 1-butanol, 2-butanona, valina, leucina, ácido lático, ácido málico, álcool, isoampilico e isoprenoides |
| EP2594648B1 (en) * | 2009-11-10 | 2016-03-16 | Massachusetts Institute of Technology | Microbial engineering for the production of chemical and pharmaceutical products from the isoprenoid pathway |
| JP5643838B2 (ja) | 2009-12-18 | 2014-12-17 | ダニスコ・ユーエス・インク | 再生可能資源からのイソプレンの精製 |
| WO2011079314A2 (en) | 2009-12-23 | 2011-06-30 | Danisco Us Inc. | Compositions and methods of pgl for the increased production of isoprene |
| EP2519629A1 (en) | 2009-12-29 | 2012-11-07 | Butamax (TM) Advanced Biofuels LLC | Expression of hexose kinase in recombinant host cells |
| US8871488B2 (en) * | 2010-06-18 | 2014-10-28 | Butamax Advanced Biofuels Llc | Recombinant host cells comprising phosphoketolases |
| EP2633043A2 (en) | 2010-10-27 | 2013-09-04 | Danisco US Inc. | Isoprene synthase variants for improved production of isoprene |
| JP5387756B2 (ja) | 2011-01-20 | 2014-01-15 | トヨタ自動車株式会社 | 組換え酵母及びこれを用いた物質生産方法 |
| WO2012149469A1 (en) * | 2011-04-29 | 2012-11-01 | Danisco Us Inc. | Production of mevalonate, isoprene, and isoprenoids using genes encoding polypeptides having thiolase, hmg-coa synthase and hmg-coa reductase enzymatic activities |
| ES2554814T3 (es) | 2011-07-12 | 2015-12-23 | Scientist Of Fortune S.A. | Microorganismo recombinante para la producción de metabolitos útiles |
| SG11201401249PA (en) | 2011-10-07 | 2014-05-29 | Danisco Us Inc | Utilization of phosphoketolase in the production of mevalonate, isoprenoid precursors, and isoprene |
| IN2014CN03791A (ja) * | 2011-11-09 | 2015-10-16 | Amyris Inc | |
| JP6595449B2 (ja) * | 2013-03-15 | 2019-10-23 | アミリス, インコーポレイテッド | アセチル補酵素a由来化合物を生産するためのホスホケトラーゼおよびホスホトランスアセチラーゼの使用 |
-
2012
- 2012-10-05 SG SG11201401249PA patent/SG11201401249PA/en unknown
- 2012-10-05 US US13/646,562 patent/US8993305B2/en active Active
- 2012-10-05 CA CA2851265A patent/CA2851265A1/en not_active Abandoned
- 2012-10-05 MX MX2014004209A patent/MX2014004209A/es unknown
- 2012-10-05 WO PCT/US2012/059136 patent/WO2013066568A1/en active Application Filing
- 2012-10-05 CN CN201280059852.7A patent/CN104053765A/zh active Pending
- 2012-10-05 JP JP2014534810A patent/JP6420147B2/ja active Active
- 2012-10-05 DK DK12775413.3T patent/DK2764084T4/da active
- 2012-10-05 AU AU2012332996A patent/AU2012332996A1/en not_active Abandoned
- 2012-10-05 BR BR112014008278A patent/BR112014008278A2/pt not_active Application Discontinuation
- 2012-10-05 EP EP17206148.3A patent/EP3339426B1/en active Active
- 2012-10-05 EP EP12775413.3A patent/EP2764084B2/en active Active
- 2012-10-05 DK DK17206148.3T patent/DK3339426T3/da active
-
2015
- 2015-02-12 HK HK15101539.9A patent/HK1201071A1/xx unknown
- 2015-02-18 US US14/625,487 patent/US9493791B2/en active Active
-
2016
- 2016-09-28 US US15/279,184 patent/US10113185B2/en active Active
-
2018
- 2018-10-11 JP JP2018192631A patent/JP6639606B2/ja active Active
Also Published As
| Publication number | Publication date |
|---|---|
| US10113185B2 (en) | 2018-10-30 |
| JP2014531904A (ja) | 2014-12-04 |
| JP6639606B2 (ja) | 2020-02-05 |
| EP3339426B1 (en) | 2022-08-24 |
| MX2014004209A (es) | 2014-08-08 |
| US20130089906A1 (en) | 2013-04-11 |
| DK2764084T4 (da) | 2021-12-06 |
| EP2764084A1 (en) | 2014-08-13 |
| AU2012332996A1 (en) | 2014-04-24 |
| HK1201071A1 (en) | 2015-08-21 |
| US8993305B2 (en) | 2015-03-31 |
| SG11201401249PA (en) | 2014-05-29 |
| EP3339426A1 (en) | 2018-06-27 |
| CN104053765A (zh) | 2014-09-17 |
| DK3339426T3 (da) | 2022-09-12 |
| JP2019030315A (ja) | 2019-02-28 |
| BR112014008278A2 (pt) | 2017-04-18 |
| US20150225744A1 (en) | 2015-08-13 |
| EP2764084B1 (en) | 2018-02-28 |
| DK2764084T3 (en) | 2018-04-09 |
| WO2013066568A1 (en) | 2013-05-10 |
| US20170198311A1 (en) | 2017-07-13 |
| EP2764084B2 (en) | 2021-11-03 |
| US9493791B2 (en) | 2016-11-15 |
| CA2851265A1 (en) | 2013-05-10 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP6639606B2 (ja) | メバロン酸、イソプレノイド前駆体、及びイソプレンの産生におけるホスホケトラーゼの使用 | |
| US10975394B2 (en) | Recombinant microorganisms for enhanced production of mevalonate, isoprene, and isoprenoids | |
| US10364443B2 (en) | Production of mevalonate, isoprene, and isoprenoids using genes encoding polypeptides having thiolase, HMG-CoA synthase and HMG-CoA reductase enzymatic activities | |
| US11371035B2 (en) | Phosphoketolases for improved production of acetyl coenzyme A-derived metabolites, isoprene, isoprenoid precursors, and isoprenoid | |
| US20130122562A1 (en) | Production of isoprene, isoprenoid precursors, and isoprenoids using acetoacetyl-coa synthase | |
| JP2016511630A (ja) | 代案の下流メバロン酸経路を使用するイソプレン、イソプレノイド、およびイソプレノイド前駆体の生成 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20151005 |
|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20151005 |
|
| A977 | Report on retrieval |
Free format text: JAPANESE INTERMEDIATE CODE: A971007 Effective date: 20160831 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20160913 |
|
| A601 | Written request for extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A601 Effective date: 20161213 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20170209 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20170704 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20171004 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20180328 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20180628 |
|
| TRDD | Decision of grant or rejection written | ||
| A01 | Written decision to grant a patent or to grant a registration (utility model) |
Free format text: JAPANESE INTERMEDIATE CODE: A01 Effective date: 20180911 |
|
| A61 | First payment of annual fees (during grant procedure) |
Free format text: JAPANESE INTERMEDIATE CODE: A61 Effective date: 20181011 |
|
| R150 | Certificate of patent or registration of utility model |
Ref document number: 6420147 Country of ref document: JP Free format text: JAPANESE INTERMEDIATE CODE: R150 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |