JP6279562B2 - 条件付きノックアウト対立遺伝子を生成するための方法および組成物 - Google Patents
条件付きノックアウト対立遺伝子を生成するための方法および組成物 Download PDFInfo
- Publication number
- JP6279562B2 JP6279562B2 JP2015517390A JP2015517390A JP6279562B2 JP 6279562 B2 JP6279562 B2 JP 6279562B2 JP 2015517390 A JP2015517390 A JP 2015517390A JP 2015517390 A JP2015517390 A JP 2015517390A JP 6279562 B2 JP6279562 B2 JP 6279562B2
- Authority
- JP
- Japan
- Prior art keywords
- sequence
- donor
- cell
- recombinase
- nuclease
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Expired - Fee Related
Links
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/10—Processes for the isolation, preparation or purification of DNA or RNA
- C12N15/102—Mutagenizing nucleic acids
-
- A—HUMAN NECESSITIES
- A01—AGRICULTURE; FORESTRY; ANIMAL HUSBANDRY; HUNTING; TRAPPING; FISHING
- A01K—ANIMAL HUSBANDRY; CARE OF BIRDS, FISHES, INSECTS; FISHING; REARING OR BREEDING ANIMALS, NOT OTHERWISE PROVIDED FOR; NEW BREEDS OF ANIMALS
- A01K67/00—Rearing or breeding animals, not otherwise provided for; New breeds of animals
- A01K67/027—New breeds of vertebrates
- A01K67/0275—Genetically modified vertebrates, e.g. transgenic
- A01K67/0276—Knockout animals
-
- A—HUMAN NECESSITIES
- A01—AGRICULTURE; FORESTRY; ANIMAL HUSBANDRY; HUNTING; TRAPPING; FISHING
- A01K—ANIMAL HUSBANDRY; CARE OF BIRDS, FISHES, INSECTS; FISHING; REARING OR BREEDING ANIMALS, NOT OTHERWISE PROVIDED FOR; NEW BREEDS OF ANIMALS
- A01K67/00—Rearing or breeding animals, not otherwise provided for; New breeds of animals
- A01K67/027—New breeds of vertebrates
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/85—Vectors or expression systems specially adapted for eukaryotic hosts for animal cells
- C12N15/8509—Vectors or expression systems specially adapted for eukaryotic hosts for animal cells for producing genetically modified animals, e.g. transgenic
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/87—Introduction of foreign genetic material using processes not otherwise provided for, e.g. co-transformation
- C12N15/90—Stable introduction of foreign DNA into chromosome
- C12N15/902—Stable introduction of foreign DNA into chromosome using homologous recombination
- C12N15/907—Stable introduction of foreign DNA into chromosome using homologous recombination in mammalian cells
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/14—Hydrolases (3)
- C12N9/16—Hydrolases (3) acting on ester bonds (3.1)
- C12N9/22—Ribonucleases RNAses, DNAses
-
- A—HUMAN NECESSITIES
- A01—AGRICULTURE; FORESTRY; ANIMAL HUSBANDRY; HUNTING; TRAPPING; FISHING
- A01K—ANIMAL HUSBANDRY; CARE OF BIRDS, FISHES, INSECTS; FISHING; REARING OR BREEDING ANIMALS, NOT OTHERWISE PROVIDED FOR; NEW BREEDS OF ANIMALS
- A01K2217/00—Genetically modified animals
- A01K2217/07—Animals genetically altered by homologous recombination
- A01K2217/075—Animals genetically altered by homologous recombination inducing loss of function, i.e. knock out
-
- A—HUMAN NECESSITIES
- A01—AGRICULTURE; FORESTRY; ANIMAL HUSBANDRY; HUNTING; TRAPPING; FISHING
- A01K—ANIMAL HUSBANDRY; CARE OF BIRDS, FISHES, INSECTS; FISHING; REARING OR BREEDING ANIMALS, NOT OTHERWISE PROVIDED FOR; NEW BREEDS OF ANIMALS
- A01K2227/00—Animals characterised by species
- A01K2227/10—Mammal
- A01K2227/105—Murine
Description
本出願は、2012年6月12日に出願した米国仮特許出願第61/658,670号の利益を主張するものであり、その開示は参照によりその全体が本明細書に組み込まれる。
本出願は、EFS−Webを介してASCII形式で提出された配列表を含み、参照することによりその全体が本明細書に組み込まれる。2013年6月12日に作製されたそのASCII複製は、P4905R1WO_PCTSequenceListing.txtと名付けられており、49,214バイトの大きさである。
1.細胞内にドナーコンストラクトを導入するステップであって、ドナーコンストラクトが、5’相同領域、5’リコンビナーゼ認識部位、ドナー配列、3’リコンビナーゼ認識部位、および3’相同領域を含み、そのドナー配列が、少なくとも1つの中立突然変異を有する標的配列を含む、ステップと、
2.細胞内に標的遺伝子内の配列を切断する配列特異的ヌクレアーゼを導入し、それによってその細胞中に条件付きノックアウト対立遺伝子を生成するステップと、を含む。
1.標的遺伝子を含む細胞内にドナーコンストラクトを導入するステップであって、ドナーコンストラクトが、5’相同領域、5’リコンビナーゼ認識部位、ドナー配列、3’リコンビナーゼ認識部位、および3’相同領域を含み、そのドナー配列が、少なくとも1つの中立突然変異を有する標的配列を含む、ステップと、
2.細胞内に配列特異的ヌクレアーゼを導入するステップであって、ヌクレアーゼが、標的遺伝子を切断する、ステップと、
3.キャリア動物内に細胞を導入し、その細胞から条件付きノックアウト動物を生成するステップと、を含む。
1.標的遺伝子を含む接合体内にドナーコンストラクトを導入するステップであって、ドナーコンストラクトが、5’相同領域、5’リコンビナーゼ認識部位、ドナー配列、3’リコンビナーゼ認識部位、および3’相同領域を含み、そのドナー配列が、少なくとも1つの中立突然変異を有する標的配列を含む、ステップと、
2.接合体内に配列特異的ヌクレアーゼを導入するステップであって、そのヌクレアーゼが標的遺伝子を切断する、ステップと、
3.キャリア動物内にその接合体を導入し、その接合体から条件付きノックアウト動物を生成するステップと、
4.5’および3’リコンビナーゼ認識部位において組換えを触媒するリコンビナーゼタンパク質をコードする導入遺伝子を有する遺伝子導入動物と、条件付きノックアウト動物を交配させ、それによりノックアウト動物を生成するステップと、を含む。
1.5’相同領域、5’リコンビナーゼ認識部位、ドナー配列、3’リコンビナーゼ認識部位、および3’相同領域を含むドナーコンストラクトであって、そのドナー配列が少なくとも1つの中立突然変異を有する標的配列を含む、ドナーコンストラクトと、
2.標的遺伝子を認識する配列特異的ヌクレアーゼと、を含む、標的遺伝子の条件付きノックアウト対立遺伝子を生成するための組成物が提供される。
この明細書を解釈するために、以下の定義が適用され、また適切な場合には、単数形で用いられる用語は複数形を含み、逆もまた同様である。以下に記載する任意の定義が、参照により本明細書に組み込まれる任意の文献と矛盾が生じる場合、以下に記載する定義が優先する。
本発明は、部分的に、リコンビナーゼ認識配列で挟んだドナー配列と組み合わせて配列特異的エンドヌクレアーゼを用いた条件付きノックアウト対立遺伝子の作製に関連付けられる技術的課題の認識および解決に関する。この過程は、かかる配列を認識および結合し、核酸分子中の二重鎖切断を誘発するエンドヌクレアーゼを用いて、染色体等の核酸分子の特異的配列を標的化することに依拠する。二重鎖切断は、誤りがちな非相同末端結合または相同的組換えのいずれかによって修復される。通過中に相同的組換えのための鋳型が提供される場合、二重鎖切断は、その提供された鋳型を用いて修復され得る。最初の二重鎖切断は、従来の相同的組換えに基づく遺伝子標的化と比較して、標的化の頻度を数桁増加させる。原理上は、二重鎖切断に近い配列に対して相同性である適切な領域で挟まれている限り、この方法を使用して修復の部位において任意の配列を挿入することができる。しかしながら、この手法は、条件付きノックアウト対立遺伝子の作製に適用する場合、ある特定の課題と関連する。条件付きノックアウト対立遺伝子は、典型的には、条件付きノックアウト対立遺伝子が非変更対立遺伝子と実質的に類似の機能性ポリペプチドを生成するが、認識配列を認識するリコンビナーゼの存在により、ある特定の時において、またはある特定の組織内において非機能的にされ得るように、遺伝子または遺伝子の部分を挟むがその機能を無傷のまま残すloxP部位等のある特定のリコンビナーゼ認識配列を含む。
本発明の種々の態様において、標的遺伝子を含む細胞中に条件付きノックアウト対立遺伝子を生成する方法が提供される。本方法は、標的遺伝子を有する細胞内に、ドナーコンストラクトと、標的遺伝子内の配列を切断するが、ドナーコンストラクトの機能を阻害しない配列特異的ヌクレアーゼとを導入し、それによって細胞中に条件付きノックアウト対立遺伝子を生成するステップを含む。本発明のこれらの、およびさらなる態様が以下に記載される。
(1)疎水性:ノルロイシン、Met、Ala、Val、Leu、Ile、
(2)中性親水性:Cys、Ser、Thr、Asn、Gln、
(3)酸性:Asp、Glu、
(4)塩基性:His、Lys、Arg、
(5)鎖配向に影響を及ぼす残基:Gly、Pro、
(6)芳香族:Trp、Tyr、Phe。
本発明のさらなる特定の態様において、標的遺伝子の条件付きノックアウト対立遺伝子を生成するための組成物が提供される。そのような組成物は、本明細書に記載されるように、5’相同領域、5’リコンビナーゼ認識部位、ドナー配列、3’リコンビナーゼ認識部位、および3’相同領域を含むドナーコンストラクトを含む。ドナー配列は、本明細書に記載されるように、少なくとも1つの中立突然変異を有する標的配列を含む。組成物は、標的遺伝子を認識する配列特異的ヌクレアーゼをさらに含む。
以下は、本発明の方法および組成物の実施例である。上で提供される一般的な説明を考慮して、様々な他の実施形態が実践されてよいことが理解される。
実施例1:C57BL/6N受精卵内へのLrp5 ZFN mRNAの前核マイクロインジェクション
表1. C57BL/6N受精卵内へのLrp5 ZFN mRNAの前核マイクロインジェクション。KO突然変異体は、1つ以上の突然変異対立遺伝子を有するマウスを含む。KO=ノックアウト。
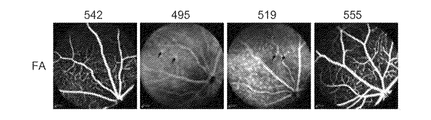
LRP5は、NORRINの共受容体として作用することによって網膜血管発達における必須の役割を果たす。NORRINシグナル伝達が妨害されると、網膜の深層における毛細血管床の形成の失敗、ならびに血管漏出に特徴付けられる血管異常を引き起こす(Xia,C.−H. et al.,Human Molecular Genetics 17,1605-1612(2008);Xia,C.−H.,PLoS ONE5,e11676(2010);Junge, H.J.et al.,Cell139,299−311(2009))。このため、実施例1に示されるように、Lrp5においてフレーム内およびフレーム外の欠失の混成を有する2月齢マウスを生成し、網膜血管発達を検査した。動物542番は、対照として働くキメラ機能性ヘテロ接合性である。この動物は、1つの野生型対立遺伝子(サイレントように見える小さい3bpのフレーム内欠失)および1bpのフレーム外欠失を有する対立遺伝子を保有した。動物495番は、4bpのフレーム外欠失対立遺伝子および1bpのフレーム外欠失対立遺伝子を含有する。動物519番は、29bpのフレーム外欠失対立遺伝子および17bpのフレーム外欠失対立遺伝子を含有する。動物555番は、3bpのフレーム内欠失対立遺伝子および1bpのフレーム外欠失対立遺伝子を有し、機能性ヘテロ接合体である。
図3Aは、エクソン2を標的とするLrp5の条件付きノックアウト対立遺伝子を生成するために用いられた戦略の概略図を示す(Gu,H.,Science 265,103−106(1994))。ZFN対は、Lrp5エクソン2に二重鎖切断を導入する(中断された矢印で示される)。切断は、ストランド侵入を介したドナープラスミドの侵入、ならびにドナープラスミドの5’と3’との間のLrp5相同領域と、エクソン2のそれぞれの相同性配列5’および3’との相同的組換えによって修復される。結果として生じる遺伝子座は、2つのloxP部位で挟まれたコドン最適化されたLrp5エクソン2を含有する(図1A、下)。
表2. Lrp5ZFN mRNA(mRNA)およびCKOドナー1プラスミドの共マイクロインジェクションKO突然変異体は、1つ以上の突然変異対立遺伝子を有するマウスを含む。KO=ノックアウト、CKO=条件付きノックアウト。a1匹のマウス(95番)は、偽陽性であった(Lrp5遺伝子座中に組み込まれたドナー1プラスミド)。bマウス140番および155番。
表3. プライマーヌクレオチド配列。プライマーP19:F=フルオロフォア(フルオレセイン)、Q=消光剤(Iowa Black FQ,Integrated DNA Technologies)、IQ=内部消光剤(ZEN,Integrated DNA Technologies)。LNA bpに下線が引かれている。
表4. Lrp5 ZFN mRNAとCKOドナー1またはドナー2プラスミドとの共マイクロインジェクション。4.5ng/μlのZFN mRNAおよび3ng/μlのドナープラスミドDNAを使用してすべての実験を実施した。全体のCKO率は、ドナー1に関しては1/12(8.3%)であり、またドナー2に関しては1/35(2.9%)であった。aマウス243番、bマウス号250番、c3’loxP部位のみの対立遺伝子を保有させた1匹のマウス(274番)、dドナー2エクソンのみを有する(loxP部位を有さない)1つの対立遺伝子ならびに1つの偽陽性対立遺伝子(Lrp5遺伝子座内に組み込まれたドナー2プラスミド)を保有させた1匹のマウス(280番)。
C57BL/6N ES細胞に、2つのLrp5 ZFN対成分をコードするプラスミドを単体で、またはマイクロインジェクション実験に用いたドナープラスミドもしくは変更されていないloxPが導入された野生型Lrp5エクソン2プラスミド(ドナー3)のいずれかと共に、エレクトロポレーションによって共トランスフェクトした。C2 ES細胞(Gertsenstein,M.et al.,PLoS ONE5,e11260(2010))を培養し、増殖し、確立された方法(Nagy,A.,Gertsenstein,M.,Vintersten,K.and Behringer,R.Manipulating the Mouse Embryo:A Laboratory Manual,Third Edition.800(Cold Spring Harbor Laboratory Press:2002))を使用して電気穿孔した。簡潔に、1500万個の細胞に、15μgのドナープラスミドを伴ってまたは伴わずに、それぞれ15μgのZFNプラスミドを電気穿孔した。培地で電気穿孔された細胞を回収し、段階希釈を支持細胞層上の10cmプレート上に平板培地した。7〜8日かけて細胞を成長させた後、各実験から144個のクローン(1.5 96ウェルプレート)を採取し、増殖のための支持細胞と共に96ウェルプレート中に播種した、。播種の2日後、細胞を支持細胞と共に新たな96ウェルプレートに1:2に分割した。次いで、一方のプレートを−80℃で保管し、他方のプレートをDNA分析のために支持細胞なしで1%のゼラチンのみと共に新たな96ウェルプレートに分割した。ES細胞を一晩溶解したことを除いて実施例1に記載されるようにDNAを単離し、DNAを沈殿し、洗浄し、翌日、基本的にRamirez−Solis,R.et al.,Anal Biochem 201,331−335(1992)に記載されるように、TE緩衝液中で再懸濁した。
表5. C57BL/6N ES細胞中への、単体またはCKOドナー1、2、または3と組み合わせてLrp5 ZFN対をコードするプラスミドのエレクトロポレーション。15μgのドナーDNAならびに/またはそれぞれ15μgのZFN1およびZFN2を使用してすべての実験を実施した。aドナー1ESクローンC8番、b5’loxPのみの対立遺伝子およびドナー2エクソンのみの(loxP部位を有さない)対立遺伝子を保有させた1つのドナー2クローン(F5)、3’loxPのみの対立遺伝子を保有させたクローンH10、c5’loxPのみを有する対立遺伝子を保有させた2つのドナー3クローン(E3およびE4)。クローンE3はまた、偽陽性対立遺伝子を保有した(ドナー3プラスミド組込み)。クローンE4はまた、真のCKOマイナー対立遺伝子(シークエンシグされた240個のTOPOクローンのうちの1陽性)を保有した。ND:調査なし、NA:該当なし。
表6. CKOドナープラスミドに由来するLrp5対立遺伝子の概要。
ドナー1(図4A、図5)から得られた条件付きノックアウト対立遺伝子におけるサイレント突然変異がLrp5遺伝子の正常な機能に影響したかどうかを決定するために、1つのノックアウト対立遺伝子(140番)および1つの条件付きノックアウト対立遺伝子(155番)を保有するマウスを実施例3のZFN対を使用して生成されたLrp5ノックアウトホモ接合性マウスと交配させた。年齢を対応させた生後16日(P16)の対照マウス(図6A、+/+)をLrp5ヘテロ接合性交配から得た。実験に使用した他のマウスは、Lrp5 KO/KO雌性とLrp5 CKO/+雄性との交配から得た。Lrp5 KO/KO雌性(図6B)は、KO/+(図6C、P16)およびCKO/KO(図6D、P16)の成体の母である。図6A−Dは、イソレクチンB4(IB4)で染色された網膜の全組織標本の代表的な共焦点投影を示す(スケールバー:50μm)。図6A−Dに示される各投影に関して、左画像は最大XY投影を示し、右画像は、神経線維層(NFL)、内網状層(IPL)、および外網状層(OPL)中の脈管構造を表すZ投影を示す(図6Dの右下パネル上に標識する)。Lrp5欠損動物は、XY投影において減少した血管複雑性ならびに深い血管層の不在(図6B)を示した。欠損背景(null background)上に条件付きノックアウト対立遺伝子を保有するマウスは、条件付きノックアウト対立遺伝子が機能性であることを示唆する正常な血管形質(図6D)を示す。図6Eは、IB4、MECA32、およびDAPIで染色された、図6A〜Dに示されるものと反対の目の網膜の断面図を示す。ホモ接合性ノックアウトマウスが有窓内皮細胞マーカーMECA32を異所的に発現したのに対して、CKO/KO、KO/+、および+/+マウスはMECA32陰性である。要約すると、ホモ接合性ノックアウト動物が上述の網膜形質(図6)を示すのに対して、1つのノックアウト対立遺伝子および1つの条件付きノックアウト対立遺伝子を保有するマウスの網膜形質は、野生型マウスまたは条件付きノックアウト対立遺伝子が機能性対立遺伝子であることを示す1つのノックアウト対立遺伝子および1つの野生型対立遺伝子のいずれかを有するマウスのものからは識別できなかった(図6)。まとめると、これらの結果は、中立突然変異を有するリコンビナーゼ認識部位で挟んだドナー配列を配列特異的ヌクレアーゼと共に用いて、インビトロおよびインビボで完全機能性条件付きノックアウト対立遺伝子を生成できることを示す。
表7. 上述の実施例で使用したPCR反応の条件
表8. 保存的置換。
本明細書に記載される方法および組成物と共に他の配列特異的エンドヌクレアーゼが使用され得るかを確認するために、Cas9/CRISPR系を用いてLrp5の変更対立遺伝子を生成した。10%FBS、L−グルタミン、および抗生物質を追加したRPMI中でHepa1−6マウス肝癌細胞を培養した。トリプシン処理およびペレット化の後、製造業者の指示に従って、AMAXA Nucleofector program T−028と共にAMAXA NucleofectorキットV(Lonza)を使用して、プラスミド1つ当たり2μgを含有するhCas9をコードするcDNAまたはCas9をコードする15μgのmRNA(図14、配列番号43)を、106細胞に電気穿孔し、6ウェルプレート中に播種した。Nucleofection効率は、GFP発現(PMAXGFP)によって評価されるように、80〜95%に達した。nucleofectionの24時間後に新鮮な培地を交換し、nucleofectionの72時間後にDNeasy Blood and Tissueキット(Qiagen)を使用して精製したゲノムDNAを収集した。ポリAテーリング反応を含む製造業者のプロトコルに従って、MMESSAGE MMachine T7 Ultraキット(Life Technologies)を用いてHCas9 mRNAをインビトロで転写した。mRNAを精製し、標準フェノール(クロロホルム抽出物およびRNAの沈殿物)を使用して濃縮した。
Hepa1−6細胞に、Cas9プラスミドまたはmRNA、gRNA、およびコドン最適化エクソン配列を含むLrp5 CKOドナー1を共トランスフェクトした。比較のため、いくつかの細胞にLrp5 ZFNプラスミドおよびドナープラスミドを共トランスフェクトした(図10)。72時間後、トランスフェクトした細胞からのゲノムDNAを、コドン最適化Lrp5ドナーエクソンに特異的なプライマー(P7;配列番号7)および3’相同性アームの外側の領域に特異的なプライマー(P12;配列番号12)を用いたPCRによって分析した。プライマーP7およびP12は、以下の条件でREDExtract−N−Amp PCR ReadyMix(Sigma)を用いたPCR反応に使用した:95℃で3分、95℃で45秒の38サイクル、63℃で45秒、72℃で1分30秒、続いて72℃で7分。PCR生成物を1%アガロースゲル上での電気泳動によって分離した。上述のように、Lrp5エクソン2ドナー1ベクターは、最初の13bpおよび最後の11bp、ならびに外因性隣接loxP部位の突然変異を除く、多くの中立突然変異を持つコドン最適化エクソン(COエクソン2)を含有する。上述のPCRは、COエクソン2配列に対して特異的な順方向プライマーおよびゲノム遺伝子座の相同性アームの外側の逆方向プライマーを使用し、ゆえに、ドナーエクソン配列が正しいLrp5遺伝子座に組み込まれた場合のみ、PCR生成物を生成する。gRNA/Cas9の使用は、ZFN系および同じドナーベクター戦略を使用するときに観察されるものを上回る優れた効率で、Lrp5遺伝子座にドナー配列組込みをもたらした(図10)。
ドナー設計戦略およびCas9/CRISPR系を用いてゲノム遺伝子座にloxP部位を導入することができるかどうか決定するために、実施例7に記載されるようにトランスフェクトされた細胞からのゲノムDNAを、相同性アームの外側に位置する1つのプライマーおよびドナーからの5’または3’loxP部位のいずれかに固定される1つのプライマーを使用するPCR分析によって分析した。5’ゲノムから5’loxPの反応に関して、2%の最終濃度へのDMSOの添加を除いて標準Expand High Fidelity PCR System(Roche)プロトコルを用いてプライマーP9およびP10(配列番号9および10)を使用した。PCRパラメータは以下の通りであった:95℃で3分、95℃で45秒の45サイクル、63℃で45秒、72℃で1分30秒、続いて72℃で7分。3’loxPから3’ゲノムの反応に関して、標準REDExtract−N−Amp PCR ReadyMix(Sigma)プロトコルに従って、プライマーP11およびP12を使用した。PCRパラメータは以下の通りであった:95℃で3分、95℃で45秒の40サイクル、62.5℃で45秒、72℃で1分30秒、続いて72℃で7分。PCR生成物を1%アガロースゲル上での電気泳動によって分離した。2つの異なるLrp5 gRNAおよびCKOドナーのうちのいずれかをトランスフェクトされた細胞から単離された試料から、3’loxP部位のためにPCR生成物を得た(図11、p_gRNA T2)。同様に、5’loxP部位に関してgRNA T7をトランスフェクトされた細胞から単離された試料からPCR生成物を得た(図11)。このため、図11は、Hepa1−6細胞において、Lrp5 gRNA T2/Cas9、およびLrp5 gRNA T7/Cas9媒介性二重鎖切断が、コドン最適化エクソンドナーベクター戦略を使用して、Lrp5遺伝子座にloxP部位の導入をもたらしたことを示す。Cas9、gRNA、およびドナーを電気穿孔された細胞のみが、ゲノム遺伝子座における5’(図11、上)および3’(図11、下)loxP部位の証拠を発現する。組み込まれたloxP部位がZFNで検出可能ではなかったのに対して、gRNA T7は、より顕著な5’loxPの存在をもたらした。Hepa1−6細胞を用いたこれらの実験において、ZFN試料における検出可能なloxP部位の不在およびgRNA試料における低レベルは、細胞株中の低い相同的組換え率およびクローンのサブセットではなく、トランスフェクトされた完全な細胞プールを分析したという事実の両方によって説明され得る。Lrp5CKO/wt遺伝子型を有する単一のマウスゲノムDNA試料を陽性対照として使用した。これらの結果は、CKO設計戦略を体細胞において使用することができ、それは、二重鎖切断と5’および3’loxP部位の両方の位置との間の望ましくない交差事象の頻度を効果的に減少させることを示す。要約すると、操作されたコドン最適化CKOドナー配列を使用してその後修復されるRNA誘導型ヌクレアーゼ媒介性DNA切断を導入することによる特異的ゲノム遺伝子座の標的化を使用して、loxP部位を挿入し、それによって条件付きノックアウト対立遺伝子を生成することができる。
他の遺伝子が本発明の方法で標的とされ得るかを確認するために、Usp10、Nnmt、およびNotch3ゲノム遺伝子座に対するドナーおよびgRNAを生成した。それぞれの遺伝子座にDNA二重鎖切断を導入し、その後、鋳型としてコドン最適化ドナーを用いて修復するために、実施例6に記載されるように、および図12において示されるように、Hepa1−6細胞にこれらのCas9/gRNAおよびドナーを導入した。基本的に上述のように、SURVEYORアッセイを実施した。プライマーP9、P12、P22、P23、P24、P25(それぞれ、配列番号9、12、22、23、24、および25)および以下のパラメータをLA Taq(Takara)と共に使用して、Lrp5、Usp10、およびNotch3ゲノム遺伝子座に対して特異的な2.2〜2.7kbの大きさのPCR生成物を増幅した:95℃で3分、95℃で45秒の35サイクル、Taで45秒(Lrp5=57C、Usp10&Notch3=63)、70℃で2分30秒、続いて72℃で7分。それぞれ、7分の1、1/3、およびすべてのPCR生成物を製造業者の指示(Transgenomic)に従ってSURVEYORアッセイにおいて使用した。野生型の鎖および突然変異対立遺伝子がアニールされたヌクレアーゼ切断を表す結果として得られる消化された生成物を1.5%アガロースゲル上での電気泳動によって分離した。
Lrp5遺伝子座を、本明細書に記載されるLrp5特異的gRNAによって標的とし、loxPが導入されたコドン最適化エクソンを導入し、それによって、条件付きノックアウト対立遺伝子を作製することができる。条件付きノックアウト対立遺伝子を持つ細胞におけるその後のCreリコンビナーゼタンパク質の発現は、ノックアウト対立遺伝子をもたらすloxPが導入されたエクソンを切除し得る。
表9. プライマーヌクレオチド配列。
Claims (28)
- 標的遺伝子を含む細胞中で条件付きノックアウト対立遺伝子を生成する方法であって、
a)前記細胞内にドナーコンストラクトを導入するステップであって、前記ドナーコンストラクトが、5’相同領域、5’リコンビナーゼ認識部位、ドナー配列、3’リコンビナーゼ認識部位、および3’相同領域を含み、前記ドナー配列は、ドナー配列が100より多い連続する非変更塩基対を含まないように、その配列の長さに沿って離れている3以上の中立突然変異を含み、ドナー配列および標的配列の間の配列相同性が90%以下である、ステップ、及び
b)前記細胞内に前記標的遺伝子内の配列を切断する配列特異的ヌクレアーゼを導入し、それによって前記細胞中に条件付きノックアウト対立遺伝子を生成するステップを含む、方法。 - 前記配列特異的ヌクレアーゼがジンクフィンガーヌクレアーゼ(ZFN)である、請求項1に記載の方法。
- 前記配列特異的ヌクレアーゼが転写活性化因子様エフェクターヌクレアーゼ(TALEN)である、請求項1に記載の方法。
- 前記配列特異的ヌクレアーゼが前記標的遺伝子を一度だけ切断するZFN二量体である、請求項1に記載の方法。
- 前記配列特異的ヌクレアーゼがRNA誘導型ヌクレアーゼである、請求項1に記載の方法。
- 前記RNA誘導型ヌクレアーゼがCas9である、請求項5に記載の方法。
- 前記配列特異的ヌクレアーゼが、タンパク質、mRNA、またはcDNAとして導入される、請求項1に記載の方法。
- 前記リコンビナーゼ認識部位が、loxP部位、frt部位、またはrox部位である、請求項1に記載の方法。
- 前記ドナー配列が7つのサイレント突然変異を含む、請求項1〜8のいずれか一項に記載の方法。
- 前記ドナー配列と前記標的配列との間の配列相同性が78%である、請求項1〜9のいずれか一項に記載の方法。
- 前記ドナーコンストラクトが、配列番号30、31、44、45、または46の配列を含む、請求項1に記載の方法。
- 前記5’相同領域が少なくとも1.1kbを含み、前記3’相同領域が少なくとも1kbを含む、請求項1〜11のいずれか一項に記載の方法。
- 前記標的遺伝子が、Lrp5、Usp10、Nnmt、およびNotch3からなる群から選択される、請求項1に記載の方法。
- 前記細胞が哺乳動物から単離されている、請求項1〜13のいずれか一項に記載の方法。
- 前記哺乳動物が、マウス、ラット、ウサギ、ハムスター、モルモット、ネコ、イヌ、ヒツジ、ウマ、ウシ、サル、およびヒトからなる群から選択される、請求項14に記載の方法。
- 前記細胞が、接合体または多能性幹細胞である、請求項1〜15のいずれか一項に記載の方法。
- 条件付きノックアウト非ヒト動物を生成する方法であって、
a)標的遺伝子を含む細胞内にドナーコンストラクトを導入するステップであって、前記ドナーコンストラクトが、5’相同領域、5’リコンビナーゼ認識部位、ドナー配列、3’リコンビナーゼ認識部位、および3’相同領域を含み、前記ドナー配列は、ドナー配列が100より多い連続する非変更塩基対を含まないように、その配列の長さに沿って離れている3以上の中立突然変異を含み、ドナー配列および標的配列の間の配列相同性が90%以下である、ステップ、
b)前記細胞内に配列特異的ヌクレアーゼを導入するステップであって、前記ヌクレアーゼが前記標的遺伝子を切断する、ステップ、及び
c)キャリア非ヒト動物内に前記細胞を導入し、前記細胞から前記条件付きノックアウト非ヒト動物を生成するステップを含む、方法。 - 前記細胞が、接合体または多能性幹細胞である、請求項17に記載の方法。
- ノックアウト非ヒト動物を生成する方法であって、
a)標的遺伝子を含む細胞内にドナーコンストラクトを導入するステップであって、前記ドナーコンストラクトが、5’相同領域、5’リコンビナーゼ認識部位、ドナー配列、3’リコンビナーゼ認識部位、および3’相同領域を含み、前記ドナー配列は、ドナー配列が100より多い連続する非変更塩基対を含まないように、その配列の長さに沿って離れている3以上の中立突然変異を含み、ドナー配列および標的配列の間の配列相同性が90%以下である、ステップ、
b)前記細胞内に配列特異的ヌクレアーゼを導入するステップであって、前記ヌクレアーゼが前記標的遺伝子を切断する、ステップ、
c)キャリア非ヒト動物内に前記細胞を導入し、前記トランスフェクトされた細胞から遺伝子導入非ヒト動物を生成するステップ、及び
d)前記5’および3’リコンビナーゼ認識部位において組換えを触媒するリコンビナーゼタンパク質をコードする導入遺伝子を有する遺伝子導入非ヒト動物と、前記条件付きノックアウト非ヒト動物を交配させるステップを含む、方法。 - 前記細胞が接合体または多能性幹細胞である、請求項19に記載の方法。
- 前記リコンビナーゼ認識部位がloxP部位であり、前記リコンビナーゼがCreリコンビナーゼである、請求項19に記載の方法。
- 前記リコンビナーゼ認識部位がfrt部位であり、前記リコンビナーゼがFLPリコンビナーゼである、請求項19に記載の方法。
- 前記リコンビナーゼ認識部位がrox部位であり、前記リコンビナーゼがDreリコンビナーゼである、請求項19に記載の方法。
- 前記リコンビナーゼをコードする前記導入遺伝子が、組織特異的プロモーターまたは誘導型プロモーターの制御下にある、請求項19に記載の方法。
- 標的遺伝子の条件付きノックアウト対立遺伝子を生成するための組成物であって、
a)5’相同領域、5’リコンビナーゼ認識部位、ドナー配列、3’リコンビナーゼ認識部位、および3’相同領域を含むドナーコンストラクトであって、前記ドナー配列は、ドナー配列が100より多い連続する非変更塩基対を含まないように、その配列の長さに沿って離れている3以上の中立突然変異を含み、ドナー配列および標的配列の間の配列相同性が90%以下である、ドナーコンストラクト、及び
b)前記標的遺伝子を認識する配列特異的ヌクレアーゼを含む、組成物。 - 前記配列特異的ヌクレアーゼが、ZFN、TALEN、およびRNA誘導型ヌクレアーゼからなる群から選択される、請求項25に記載の組成物。
- 配列番号30、31、44、45、または46の配列を含む、ドナーコンストラクト。
- 請求項27に記載のドナーコンストラクトを含む、細胞。
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201261658670P | 2012-06-12 | 2012-06-12 | |
| US61/658,670 | 2012-06-12 | ||
| PCT/US2013/045382 WO2013188522A2 (en) | 2012-06-12 | 2013-06-12 | Methods and compositions for generating conditional knock-out alleles |
Publications (3)
| Publication Number | Publication Date |
|---|---|
| JP2015519082A JP2015519082A (ja) | 2015-07-09 |
| JP2015519082A5 JP2015519082A5 (ja) | 2017-09-28 |
| JP6279562B2 true JP6279562B2 (ja) | 2018-02-14 |
Family
ID=49758880
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2015517390A Expired - Fee Related JP6279562B2 (ja) | 2012-06-12 | 2013-06-12 | 条件付きノックアウト対立遺伝子を生成するための方法および組成物 |
Country Status (11)
| Country | Link |
|---|---|
| US (1) | US20150128300A1 (ja) |
| EP (1) | EP2858486A4 (ja) |
| JP (1) | JP6279562B2 (ja) |
| KR (1) | KR20150023670A (ja) |
| CN (1) | CN104540382A (ja) |
| BR (1) | BR112014031080A2 (ja) |
| CA (1) | CA2876076A1 (ja) |
| HK (1) | HK1209276A1 (ja) |
| MX (1) | MX2014015204A (ja) |
| RU (1) | RU2014153918A (ja) |
| WO (1) | WO2013188522A2 (ja) |
Families Citing this family (77)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US10323236B2 (en) | 2011-07-22 | 2019-06-18 | President And Fellows Of Harvard College | Evaluation and improvement of nuclease cleavage specificity |
| GB201122458D0 (en) | 2011-12-30 | 2012-02-08 | Univ Wageningen | Modified cascade ribonucleoproteins and uses thereof |
| DK2847335T3 (en) | 2012-04-25 | 2018-08-13 | Regeneron Pharma | NUCLEASED MEDIUM TARGETING WITH LARGE TARGET VECTORS |
| US10266850B2 (en) | 2012-05-25 | 2019-04-23 | The Regents Of The University Of California | Methods and compositions for RNA-directed target DNA modification and for RNA-directed modulation of transcription |
| KR102052286B1 (ko) | 2012-10-23 | 2019-12-06 | 주식회사 툴젠 | 표적 DNA에 특이적인 가이드 RNA 및 Cas 단백질을 암호화하는 핵산 또는 Cas 단백질을 포함하는, 표적 DNA를 절단하기 위한 조성물 및 이의 용도 |
| DK3138912T3 (en) | 2012-12-06 | 2019-01-21 | Sigma Aldrich Co Llc | CRISPR-BASED RE-MODIFICATION AND REGULATION |
| JP2016507244A (ja) * | 2013-02-27 | 2016-03-10 | ヘルムホルツ・ツェントルム・ミュンヒェン・ドイチェス・フォルシュンクスツェントルム・フューア・ゲズントハイト・ウント・ウムベルト(ゲーエムベーハー)Helmholtz Zentrum MuenchenDeutsches Forschungszentrum fuer Gesundheit und Umwelt (GmbH) | Cas9ヌクレアーゼによる卵母細胞における遺伝子編集 |
| JP2016519652A (ja) | 2013-03-14 | 2016-07-07 | カリブー・バイオサイエンシーズ・インコーポレイテッド | 核酸ターゲティング核酸の組成物および方法 |
| ES2888250T3 (es) | 2013-04-16 | 2022-01-03 | Regeneron Pharma | Modificación direccionada del genoma de rata |
| EP3663405A1 (en) | 2013-06-11 | 2020-06-10 | Takara Bio USA, Inc. | Protein enriched microvesicles and methods of making and using the same |
| US20150044192A1 (en) | 2013-08-09 | 2015-02-12 | President And Fellows Of Harvard College | Methods for identifying a target site of a cas9 nuclease |
| US9359599B2 (en) | 2013-08-22 | 2016-06-07 | President And Fellows Of Harvard College | Engineered transcription activator-like effector (TALE) domains and uses thereof |
| US9228207B2 (en) | 2013-09-06 | 2016-01-05 | President And Fellows Of Harvard College | Switchable gRNAs comprising aptamers |
| US9322037B2 (en) | 2013-09-06 | 2016-04-26 | President And Fellows Of Harvard College | Cas9-FokI fusion proteins and uses thereof |
| US9737604B2 (en) | 2013-09-06 | 2017-08-22 | President And Fellows Of Harvard College | Use of cationic lipids to deliver CAS9 |
| EP3988649A1 (en) | 2013-09-18 | 2022-04-27 | Kymab Limited | Methods, cells and organisms |
| WO2015066119A1 (en) | 2013-10-30 | 2015-05-07 | North Carolina State University | Compositions and methods related to a type-ii crispr-cas system in lactobacillus buchneri |
| LT3066201T (lt) | 2013-11-07 | 2018-08-10 | Editas Medicine, Inc. | Su crispr susiję būdai ir kompozicijos su valdančiomis grnr |
| SG10201700961TA (en) | 2013-12-11 | 2017-04-27 | Regeneron Pharma | Methods and compositions for the targeted modification of a genome |
| US20150166984A1 (en) | 2013-12-12 | 2015-06-18 | President And Fellows Of Harvard College | Methods for correcting alpha-antitrypsin point mutations |
| CN103725712B (zh) * | 2014-01-17 | 2016-03-30 | 上海金卫生物技术有限公司 | 一种无物种限制的条件性基因敲除用中间载体及其制备方法和用途 |
| US10787654B2 (en) | 2014-01-24 | 2020-09-29 | North Carolina State University | Methods and compositions for sequence guiding Cas9 targeting |
| CA2944978C (en) | 2014-04-08 | 2024-02-13 | North Carolina State University | Methods and compositions for rna-directed repression of transcription using crispr-associated genes |
| ES2962509T3 (es) * | 2014-04-14 | 2024-03-19 | Maxcyte Inc | Métodos y composiciones para modificar ADN genómico |
| WO2015191911A2 (en) * | 2014-06-12 | 2015-12-17 | Clontech Laboratories, Inc. | Protein enriched microvesicles and methods of making and using the same |
| EP3169776A4 (en) * | 2014-07-14 | 2018-07-04 | The Regents of The University of California | Crispr/cas transcriptional modulation |
| EP3177718B1 (en) | 2014-07-30 | 2022-03-16 | President and Fellows of Harvard College | Cas9 proteins including ligand-dependent inteins |
| CN107429241A (zh) | 2014-08-14 | 2017-12-01 | 北京百奥赛图基因生物技术有限公司 | Dna敲入系统 |
| CN107075546B (zh) * | 2014-08-19 | 2021-08-31 | 哈佛学院董事及会员团体 | 用于对核酸探测并作图的rna-引导的系统 |
| WO2016033298A1 (en) | 2014-08-28 | 2016-03-03 | North Carolina State University | Novel cas9 proteins and guiding features for dna targeting and genome editing |
| CN104293833B (zh) * | 2014-10-09 | 2017-10-10 | 西北农林科技大学 | 一种基于TALEN介导的Sp110巨噬细胞特异打靶载体及重组细胞 |
| CN104404036B (zh) * | 2014-11-03 | 2017-12-01 | 赛业(苏州)生物科技有限公司 | 基于CRISPR/Cas9技术的条件性基因敲除方法 |
| US11680268B2 (en) | 2014-11-07 | 2023-06-20 | Editas Medicine, Inc. | Methods for improving CRISPR/Cas-mediated genome-editing |
| US11470826B2 (en) | 2014-11-17 | 2022-10-18 | National University Corporation Tokyo Medical And Dental University | Method of conveniently producing genetically modified non-human mammal with high efficiency |
| AU2015349692B2 (en) | 2014-11-21 | 2021-10-28 | Regeneron Pharmaceuticals, Inc. | Methods and compositions for targeted genetic modification using paired guide RNAs |
| ES2926467T3 (es) | 2015-04-13 | 2022-10-26 | Maxcyte Inc | Métodos y composiciones para la modificación del ADN genómico |
| EP3303607A4 (en) | 2015-05-29 | 2018-10-10 | North Carolina State University | Methods for screening bacteria, archaea, algae, and yeast using crispr nucleic acids |
| JP7051438B2 (ja) | 2015-06-15 | 2022-04-11 | ノース カロライナ ステート ユニバーシティ | 核酸およびrnaに基づく抗菌剤の効率的な送達のための方法および組成物 |
| US11279928B2 (en) | 2015-06-29 | 2022-03-22 | Massachusetts Institute Of Technology | Compositions comprising nucleic acids and methods of using the same |
| JP6713691B2 (ja) * | 2015-08-20 | 2020-07-01 | 大学共同利用機関法人情報・システム研究機構 | 動物細胞ゲノム部位特異的外来dna挿入方法及び前記挿入方法を用いて得られる細胞 |
| WO2017053879A1 (en) | 2015-09-24 | 2017-03-30 | Editas Medicine, Inc. | Use of exonucleases to improve crispr/cas-mediated genome editing |
| US11286480B2 (en) | 2015-09-28 | 2022-03-29 | North Carolina State University | Methods and compositions for sequence specific antimicrobials |
| WO2017070632A2 (en) | 2015-10-23 | 2017-04-27 | President And Fellows Of Harvard College | Nucleobase editors and uses thereof |
| WO2017100343A1 (en) * | 2015-12-07 | 2017-06-15 | Arc Bio, Llc | Methods and compositions for the making and using of guide nucleic acids |
| US11542466B2 (en) | 2015-12-22 | 2023-01-03 | North Carolina State University | Methods and compositions for delivery of CRISPR based antimicrobials |
| CN107022572A (zh) * | 2016-02-01 | 2017-08-08 | 河北伊维沃生物科技有限公司 | Ldl受体基因敲除的基因工程仓鼠 |
| WO2017136794A1 (en) | 2016-02-03 | 2017-08-10 | Massachusetts Institute Of Technology | Structure-guided chemical modification of guide rna and its applications |
| KR20180110144A (ko) * | 2016-02-26 | 2018-10-08 | 란자테크 뉴질랜드 리미티드 | C1-고정 박테리아에 대한 crispr/cas 시스템 |
| US11597924B2 (en) | 2016-03-25 | 2023-03-07 | Editas Medicine, Inc. | Genome editing systems comprising repair-modulating enzyme molecules and methods of their use |
| US11236313B2 (en) | 2016-04-13 | 2022-02-01 | Editas Medicine, Inc. | Cas9 fusion molecules, gene editing systems, and methods of use thereof |
| MX2018016307A (es) * | 2016-06-27 | 2019-05-20 | Baylor College Medicine | Animal no humano quimerico con higado humano con deficiencia en p450 oxidoreductasa y metodos de uso el mismo. |
| KR20230095129A (ko) | 2016-08-03 | 2023-06-28 | 프레지던트 앤드 펠로우즈 오브 하바드 칼리지 | 아데노신 핵염기 편집제 및 그의 용도 |
| US11661590B2 (en) | 2016-08-09 | 2023-05-30 | President And Fellows Of Harvard College | Programmable CAS9-recombinase fusion proteins and uses thereof |
| WO2018039438A1 (en) | 2016-08-24 | 2018-03-01 | President And Fellows Of Harvard College | Incorporation of unnatural amino acids into proteins using base editing |
| CN107779462B (zh) * | 2016-08-29 | 2021-06-04 | 中国科学院分子细胞科学卓越创新中心 | 双同源重组谱系示踪技术 |
| US11306324B2 (en) | 2016-10-14 | 2022-04-19 | President And Fellows Of Harvard College | AAV delivery of nucleobase editors |
| GB201617559D0 (en) | 2016-10-17 | 2016-11-30 | University Court Of The University Of Edinburgh The | Swine comprising modified cd163 and associated methods |
| WO2018144097A1 (en) | 2016-11-04 | 2018-08-09 | Akeagen Llc | Genetically modified non-human animals and methods for producing heavy chain-only antibodies |
| WO2018097257A1 (ja) * | 2016-11-28 | 2018-05-31 | 国立大学法人大阪大学 | ゲノム編集方法 |
| WO2018119359A1 (en) | 2016-12-23 | 2018-06-28 | President And Fellows Of Harvard College | Editing of ccr5 receptor gene to protect against hiv infection |
| TW201839136A (zh) | 2017-02-06 | 2018-11-01 | 瑞士商諾華公司 | 治療血色素異常症之組合物及方法 |
| EP3592853A1 (en) | 2017-03-09 | 2020-01-15 | President and Fellows of Harvard College | Suppression of pain by gene editing |
| KR20190127797A (ko) | 2017-03-10 | 2019-11-13 | 프레지던트 앤드 펠로우즈 오브 하바드 칼리지 | 시토신에서 구아닌으로의 염기 편집제 |
| US11268082B2 (en) | 2017-03-23 | 2022-03-08 | President And Fellows Of Harvard College | Nucleobase editors comprising nucleic acid programmable DNA binding proteins |
| US11560566B2 (en) | 2017-05-12 | 2023-01-24 | President And Fellows Of Harvard College | Aptazyme-embedded guide RNAs for use with CRISPR-Cas9 in genome editing and transcriptional activation |
| WO2019014564A1 (en) * | 2017-07-14 | 2019-01-17 | Editas Medicine, Inc. | SYSTEMS AND METHODS OF TARGETED INTEGRATION AND GENOME EDITING AND DETECTION THEREOF WITH INTEGRATED PRIMING SITES |
| EP3658573A1 (en) | 2017-07-28 | 2020-06-03 | President and Fellows of Harvard College | Methods and compositions for evolving base editors using phage-assisted continuous evolution (pace) |
| EP3676376A2 (en) | 2017-08-30 | 2020-07-08 | President and Fellows of Harvard College | High efficiency base editors comprising gam |
| US11795443B2 (en) | 2017-10-16 | 2023-10-24 | The Broad Institute, Inc. | Uses of adenosine base editors |
| CN108998483B (zh) * | 2018-08-17 | 2021-04-30 | 武汉核圣生物技术有限公司 | 利用单亚基RNA聚合酶进行体外转录合成sgRNA的方法 |
| US10711267B2 (en) | 2018-10-01 | 2020-07-14 | North Carolina State University | Recombinant type I CRISPR-Cas system |
| CN113891937A (zh) | 2019-03-19 | 2022-01-04 | 布罗德研究所股份有限公司 | 编辑核苷酸序列的方法和组合物 |
| CN110512005B (zh) * | 2019-06-05 | 2021-05-14 | 浙江大学 | 泛素特异性蛋白酶8在调控湖羊肉质及育种中的应用 |
| IL297761A (en) | 2020-05-08 | 2022-12-01 | Broad Inst Inc | Methods and compositions for simultaneously editing two helices of a designated double-helix nucleotide sequence |
| CN113678789A (zh) * | 2021-08-26 | 2021-11-23 | 嘉兴学院 | Mir-379/410基因簇敲除的小鼠模型及其构建方法 |
| GB202118058D0 (en) | 2021-12-14 | 2022-01-26 | Univ Warwick | Methods to increase yields in crops |
| GB2621813A (en) | 2022-06-30 | 2024-02-28 | Univ Newcastle | Preventing disease recurrence in Mitochondrial replacement therapy |
Family Cites Families (17)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US713608A (en) | 1901-07-06 | 1902-11-18 | William J Condlon | Course-indicator for ships. |
| US5436150A (en) | 1992-04-03 | 1995-07-25 | The Johns Hopkins University | Functional domains in flavobacterium okeanokoities (foki) restriction endonuclease |
| EP0682699B1 (en) | 1993-02-12 | 2003-05-07 | The Johns-Hopkins University | FUNCTIONAL DOMAINS IN $i(FLAVOBACTERIUM OKEANOKOITES) (FOKI) RESTRICTION ENDONUCLEASE |
| US7135608B1 (en) | 1997-08-28 | 2006-11-14 | The Salk Institute For Biological Studies | Site-specific recombination in eukaryotes and constructs useful therefor |
| US6534261B1 (en) | 1999-01-12 | 2003-03-18 | Sangamo Biosciences, Inc. | Regulation of endogenous gene expression in cells using zinc finger proteins |
| BR0209564A (pt) * | 2001-05-11 | 2004-10-26 | Wyeth Corp | Animal transgênico, embrião, camundongo transgênico, modelo animal para o estudo da modulação da densidade óssea, célula isolada, ácido nucleico para marcar o gene pela recombinação homóloga, e, métodos para estudar determinantes da massa óssea, para estudar moduladores de massa óssea, para estudar a massa óssea, para estudar um efeito hbm sobre os distúrbios ósseos, para identificar marcadores substitutos da formação/reabsorção ósseas, para estudar efeitos de hbm sobre os distúrbios cardìacos, para avaliar tratamentos cárdio-protetores quanto aos efeitos de modulação da massa óssea, para modular a densidade óssea, para produzir um camundongo transgênico e para identificar genes associados com a massa óssea |
| US20070218071A1 (en) * | 2003-09-15 | 2007-09-20 | Morris David W | Novel therapeutic targets in cancer |
| CN101273141B (zh) * | 2005-07-26 | 2013-03-27 | 桑格摩生物科学股份有限公司 | 外源核酸序列的靶向整合和表达 |
| SG163558A1 (en) * | 2006-12-29 | 2010-08-30 | Osprey Pharmaceuticals Usa Inc | Methods of selecting and producing modified toxins, conjugates containing modified toxins and uses thereof |
| PT2336329E (pt) * | 2007-06-01 | 2012-12-24 | Omt Inc | Composições e métodos para inibição de genes de imunoglobulina endógena e produção de anticorpos transgénicos humanos idiotípicos |
| US8936936B2 (en) * | 2007-10-25 | 2015-01-20 | Sangamo Biosciences, Inc. | Methods and compositions for targeted integration |
| US20110023143A1 (en) * | 2008-12-04 | 2011-01-27 | Sigma-Aldrich Co. | Genomic editing of neurodevelopmental genes in animals |
| US20120192298A1 (en) * | 2009-07-24 | 2012-07-26 | Sigma Aldrich Co. Llc | Method for genome editing |
| KR20120097483A (ko) * | 2009-07-24 | 2012-09-04 | 시그마-알드리치 컴퍼니., 엘엘씨 | 게놈 편집을 위한 방법 |
| EP2510096B2 (en) | 2009-12-10 | 2018-02-07 | Regents of the University of Minnesota | Tal effector-mediated dna modification |
| US20120204282A1 (en) * | 2011-02-04 | 2012-08-09 | Sangamo Biosciences, Inc. | Methods and compositions for treating occular disorders |
| US10266850B2 (en) * | 2012-05-25 | 2019-04-23 | The Regents Of The University Of California | Methods and compositions for RNA-directed target DNA modification and for RNA-directed modulation of transcription |
-
2013
- 2013-06-12 JP JP2015517390A patent/JP6279562B2/ja not_active Expired - Fee Related
- 2013-06-12 CA CA 2876076 patent/CA2876076A1/en not_active Abandoned
- 2013-06-12 CN CN201380042657.8A patent/CN104540382A/zh active Pending
- 2013-06-12 US US14/407,869 patent/US20150128300A1/en not_active Abandoned
- 2013-06-12 KR KR1020157000353A patent/KR20150023670A/ko not_active Application Discontinuation
- 2013-06-12 BR BR112014031080A patent/BR112014031080A2/pt not_active IP Right Cessation
- 2013-06-12 EP EP13804001.9A patent/EP2858486A4/en not_active Withdrawn
- 2013-06-12 MX MX2014015204A patent/MX2014015204A/es unknown
- 2013-06-12 RU RU2014153918A patent/RU2014153918A/ru not_active Application Discontinuation
- 2013-06-12 WO PCT/US2013/045382 patent/WO2013188522A2/en active Application Filing
-
2015
- 2015-10-20 HK HK15110302.5A patent/HK1209276A1/xx unknown
Also Published As
| Publication number | Publication date |
|---|---|
| KR20150023670A (ko) | 2015-03-05 |
| CA2876076A1 (en) | 2013-12-19 |
| CN104540382A (zh) | 2015-04-22 |
| BR112014031080A2 (pt) | 2018-05-08 |
| JP2015519082A (ja) | 2015-07-09 |
| HK1209276A1 (en) | 2016-04-01 |
| MX2014015204A (es) | 2015-08-07 |
| RU2014153918A (ru) | 2016-07-27 |
| EP2858486A2 (en) | 2015-04-15 |
| WO2013188522A2 (en) | 2013-12-19 |
| WO2013188522A3 (en) | 2014-04-10 |
| EP2858486A4 (en) | 2016-04-13 |
| US20150128300A1 (en) | 2015-05-07 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP6279562B2 (ja) | 条件付きノックアウト対立遺伝子を生成するための方法および組成物 | |
| US11555208B2 (en) | DNA editing using relatively long single-stranded DNA and CRISPR/Cas9 to increase success rate in methods for preparing transgenic embryos and animals | |
| JP6700306B2 (ja) | 受精前の卵細胞、受精卵、及び標的遺伝子の改変方法 | |
| EP2392208B1 (en) | Fusion proteins comprising a DNA-binding domain of a Tal effector protein and a non-specific cleavage domain of a restriction nuclease and their use | |
| JP5320546B2 (ja) | Tol1因子のトランスポザーゼ及びそれを用いたDNA導入システム | |
| Li et al. | One-step efficient generation of dual-function conditional knockout and geno-tagging alleles in zebrafish | |
| Tasic et al. | Extensions of MADM (mosaic analysis with double markers) in mice | |
| EP2493288B1 (en) | Homologous recombination in the oocyte | |
| JP2018532415A (ja) | 大きなゲノムdnaノックインおよびその使用 | |
| JP7257062B2 (ja) | ゲノム編集方法 | |
| US20150156996A1 (en) | Livestock with genetically modified prolactin receptor | |
| Lee et al. | Conditional targeting of Ispd using paired Cas9 nickase and a single DNA template in mice | |
| JP6958917B2 (ja) | 遺伝子ノックイン細胞の作製方法 | |
| Takasu et al. | Precise genome editing in the silkworm Bombyx mori using TALENs and ds-and ssDNA donors–A practical approach | |
| US20120124686A1 (en) | Site-Directed Integration of Transgenes in Mammals | |
| US20220369610A1 (en) | High frequency targeted animal transgenesis | |
| US20040068761A1 (en) | Ends-out gene targeting method | |
| Shi et al. | Targeted integration of genes in Xenopus tropicalis | |
| Wierson | Using zebrafish to iterate and expand the precision genome writing toolbox | |
| US20210189404A1 (en) | Method for producing fusion protein, nucleic acid, cell, and animal | |
| De Cian et al. | Efficient gene targeting by homology-directed repair in rat zygotes using TALE nucleases |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20160418 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20170307 |
|
| A601 | Written request for extension of time |
Free format text: JAPANESE INTERMEDIATE CODE: A601 Effective date: 20170601 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20170807 |
|
| A524 | Written submission of copy of amendment under article 19 pct |
Free format text: JAPANESE INTERMEDIATE CODE: A524 Effective date: 20170807 |
|
| TRDD | Decision of grant or rejection written | ||
| A01 | Written decision to grant a patent or to grant a registration (utility model) |
Free format text: JAPANESE INTERMEDIATE CODE: A01 Effective date: 20171219 |
|
| A61 | First payment of annual fees (during grant procedure) |
Free format text: JAPANESE INTERMEDIATE CODE: A61 Effective date: 20180117 |
|
| R150 | Certificate of patent or registration of utility model |
Ref document number: 6279562 Country of ref document: JP Free format text: JAPANESE INTERMEDIATE CODE: R150 |
|
| LAPS | Cancellation because of no payment of annual fees |