WO2011021508A1 - C型肝炎の治療効果を予測するためのマーカー及びc型肝炎の治療効果の予測を行う方法並びにc型肝炎の予防又は治療剤 - Google Patents
C型肝炎の治療効果を予測するためのマーカー及びc型肝炎の治療効果の予測を行う方法並びにc型肝炎の予防又は治療剤 Download PDFInfo
- Publication number
- WO2011021508A1 WO2011021508A1 PCT/JP2010/063262 JP2010063262W WO2011021508A1 WO 2011021508 A1 WO2011021508 A1 WO 2011021508A1 JP 2010063262 W JP2010063262 W JP 2010063262W WO 2011021508 A1 WO2011021508 A1 WO 2011021508A1
- Authority
- WO
- WIPO (PCT)
- Prior art keywords
- hepatitis
- therapeutic effect
- polynucleotide
- predicting
- marker
- Prior art date
Links
Images
Classifications
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K31/00—Medicinal preparations containing organic active ingredients
- A61K31/70—Carbohydrates; Sugars; Derivatives thereof
- A61K31/7088—Compounds having three or more nucleosides or nucleotides
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K38/00—Medicinal preparations containing peptides
- A61K38/16—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- A61K38/17—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- A61K38/19—Cytokines; Lymphokines; Interferons
- A61K38/21—Interferons [IFN]
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P1/00—Drugs for disorders of the alimentary tract or the digestive system
- A61P1/16—Drugs for disorders of the alimentary tract or the digestive system for liver or gallbladder disorders, e.g. hepatoprotective agents, cholagogues, litholytics
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P31/00—Antiinfectives, i.e. antibiotics, antiseptics, chemotherapeutics
- A61P31/12—Antivirals
- A61P31/14—Antivirals for RNA viruses
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N7/00—Viruses; Bacteriophages; Compositions thereof; Preparation or purification thereof
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6876—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes
- C12Q1/6883—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes for diseases caused by alterations of genetic material
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2770/00—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA ssRNA viruses positive-sense
- C12N2770/00011—Details
- C12N2770/24011—Flaviviridae
- C12N2770/24211—Hepacivirus, e.g. hepatitis C virus, hepatitis G virus
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/106—Pharmacogenomics, i.e. genetic variability in individual responses to drugs and drug metabolism
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/156—Polymorphic or mutational markers
Definitions
- the present invention comprehensively verifies the entire host genome, predicts the therapeutic effect of hepatitis C, and more accurately predicts the therapeutic effect of hepatitis C using the marker.
- the present invention relates to a method, a probe used in the prediction, a primer, or a kit containing them, and a prophylactic or therapeutic agent for hepatitis C.
- IFN interferon
- the present inventors conducted an SNP analysis of “PEGylated IFN / RBV combination therapy” comprehensively over the entire host genome in an Asian population centered on Japanese ( As a result of Genome-Wide Association Study (GWAS), we found many SNPs with a high risk rate (it is difficult to obtain a therapeutic effect) and found gene regions where the SNPs were concentrated.
- the purpose is to use a highly accurate marker for predicting the therapeutic effect of hepatitis C using SNP and other gene polymorphisms, a probe, primer or kit used for the prediction, and C using the marker.
- Method for predicting the therapeutic effect of hepatitis B, and a gene for searching for a marker for predicting the therapeutic effect, as well as prevention of hepatitis C using the protein encoded by the region where the SNP of the present invention and other gene polymorphisms are concentrated Is to provide a therapeutic agent.
- Oligonucleotides or polynucleotides present on the human genome including one or more selected from the following polymorphisms or those in linkage disequilibrium (R2> 0.8) A marker for predicting the therapeutic effect of hepatitis C.
- ⁇ Second invention> The marker for predicting the therapeutic effect of hepatitis C according to the first invention, characterized in that the length of the oligonucleotide or polynucleotide is 40 k bases or less.
- ⁇ Third invention> The therapeutic effect of hepatitis C according to the first invention or the second invention, wherein the therapeutic effect of hepatitis C is a therapeutic effect by treatment with interferon and / or PEGylated interferon A marker for prediction.
- ⁇ Fourth Invention> The therapeutic effect of hepatitis C according to any one of the first to third inventions, wherein the therapeutic effect of hepatitis C is a therapeutic effect of PEGylated interferon / RBV combination therapy Marker to do.
- ⁇ Fifth invention Oligonucleotide or polynucleotide present on the human genome, which hybridizes to one or more selected from the gene polymorphisms described in the first invention, or a label thereof object.
- ⁇ Sixth invention> A probe comprising the oligonucleotide or polynucleotide according to the fifth invention, or a label thereof.
- a primer comprising the oligonucleotide or polynucleotide according to the fifth invention, or a label thereof.
- ⁇ Eighth invention> A kit for predicting the therapeutic effect of hepatitis C, comprising the probe according to the sixth invention and / or the primer according to the seventh invention.
- ⁇ Ninth Invention> A method for predicting the therapeutic effect of hepatitis C, comprising the following steps (1) and (2).
- (1) The process of determining the genotype of the following (X) with the sample obtained from the patient.
- (X) A step of predicting the therapeutic effect of hepatitis C from the genotype determined by at least one of the gene polymorphisms described in the first invention (2) (1).
- ⁇ Tenth invention> The method for predicting the therapeutic effect of hepatitis C according to the ninth invention, wherein the therapeutic effect of hepatitis C is a therapeutic effect by treatment with interferon and / or PEGylated interferon.
- ⁇ Eleventh invention> The method for predicting the therapeutic effect of hepatitis C according to the tenth invention, wherein the therapeutic effect of hepatitis C is a therapeutic effect of PEGylated interferon / RBV combination therapy.
- a gene for searching for a therapeutic effect prediction marker for hepatitis C which has the following sequence (A) or (B): (A) Oligonucleotide or polynucleotide (B) (A) comprising at least a part of the polynucleotide represented by SEQ ID NO: 59, wherein one to several nucleotides are deleted, substituted, added, and / or Inserted oligonucleotide or polynucleotide
- a screening method for a therapeutic agent for hepatitis C wherein at least one of the following (A) to (C) is used.
- (A) Oligonucleotide or polynucleotide (B) (A) comprising at least a part of the polynucleotide represented by SEQ ID NO: 59, wherein one to several nucleotides are deleted, substituted, added, and / or Inserted oligonucleotide or polynucleotide (C) oligonucleotide or polynucleotide present on the human genome, including rs8103142
- ⁇ 14th invention A prophylactic or therapeutic agent for hepatitis C, comprising IL28B protein and / or a gene encoding it.
- the “marker for predicting the therapeutic effect of hepatitis C” of the present invention is based on the result of exhaustive verification over the entire host genome, the treatment of hepatitis C, particularly interferon therapy, especially the latest treatment It is an index that can more accurately predict the therapeutic effect of a certain “PEGylated IFN / RBV combination therapy”. Also, the risk value is clearer than the conventional marker (P value is considerably smaller), and judgment is easy.
- the therapeutic effect of interferon therapy especially “PEGylated IFN / RBV combination therapy”, which is the latest treatment, can be predicted more accurately.
- a new marker for predicting the therapeutic effect of hepatitis C can be discovered more quickly.
- the “method for screening a therapeutic agent for hepatitis C” of the present invention it is possible to screen for therapeutic agents for hepatitis C other than interferon and RBV. With the preventive or therapeutic agent of the present invention, effective prevention or treatment of hepatitis C can be expected.
- FIG. 3 is a diagram (continuation of FIG. 2) showing the positions of two SNP positions (major alleles) in a part of the polynucleotide represented by SEQ ID NO: 59.
- “gene”, “oligonucleotide”, and “polynucleotide” are purine bases such as adenine (A) and guanine (G), thymine (T), uracil (U), cytosine ( C) and other pyrimidine bases and their modified bases as constituents, from single-stranded or double-stranded DNA, single-stranded or double-stranded RNA, single-stranded DNA and single-stranded RNA Or any one or a plurality of chimeras in which RNA and DNA are combined to form a single strand.
- DNA also includes cDNA.
- the “marker for predicting the therapeutic effect of hepatitis C” of the present invention is an index for predicting the therapeutic effect of hepatitis C, and is an oligonucleotide or a polynucleotide present on the human genome. And one or more selected from polymorphisms shown below, or those in linkage disequilibrium (R2> 0.8).
- the specific gene polymorphism and “in a linkage disequilibrium state (R2> 0.8)” can predict the therapeutic effect similar to the specific gene polymorphism.
- Haploview software or the like it can be detected according to known methods.
- the marker is a gene or gene fragment present in a sample of blood or the like of an infected person with hepatitis C or a subject suspected of being infected, and is detected by a probe or the like described later, By examining the genotype (gene polymorphism such as SNP), it becomes a sample for predicting the therapeutic effect of hepatitis C.
- genotype gene polymorphism such as SNP
- SEQ ID NO: 59 is an example of a sequence including a gene encoding IL28B present in the human genome and its periphery.
- IL28B is a gene on the so-called antisense strand in two chromosomes.
- alleles are expressed by chromosome sense strands. Therefore, the description of major alleles of the four SNPs (rs11881222, rs8103142, rs28416813, rs4803219) present in SEQ ID NO: 59 (sites surrounded by the frames in FIGS. 2 and 3) are complementary to the major alleles shown in Table 3. It has become.
- the above-mentioned “gene polymorphism” means an unusual genotype having a frequency of 1% or more of the population, and lacking not only SNP but also one to several nucleotides. In addition to mutations such as loss, substitution, addition, and / or insertion, it also includes VNTR (variable number of tandem : repeat), STRP (short tandem repeat polymorphism), etc. It includes those in which a part of the repetitive polymorphism has undergone mutation such as deletion, substitution, addition, and / or insertion.
- Such gene polymorphisms other than SNP are also included in the marker of the present invention because there may be a deletion of several nucleotides in the promoter region of the polynucleotide represented by SEQ ID NO: 59, This deletion is considered to be highly likely to be a genetic polymorphism associated with resistance to hepatitis C treatment, as it is linked to the risk allele of some of the SNPs described above. is there. Specifically, “TATA repeat sequence (positions ⁇ 595 to ⁇ 568)” present in the promoter region upstream of the start codon of the IL28B gene present in SEQ ID NO: 59 (300 in FIG.
- the “gene polymorphism present in the polynucleotide represented by SEQ ID NO: 59” means “the SNP listed above among various gene polymorphisms present in the polynucleotide represented by SEQ ID NO: 59”. And other known or unknown SNPs and other gene polymorphisms.
- Such gene polymorphisms are included because many of the SNPs described above are present in the polynucleotide region of SEQ ID NO: 59, and therefore SEQ ID NO: 59 itself may be involved in the therapeutic effect of hepatitis C. This is because if the gene polymorphism in this region is high, the possibility of being involved in predicting the therapeutic effect of hepatitis C is considered high.
- the marker of the present invention including “a gene polymorphism present in the polynucleotide represented by SEQ ID NO: 59” includes (indicating a gene polymorphism) among the polynucleotides represented by SEQ ID NO: 59. Also included are oligonucleotides or polynucleotides in which one to several nucleotides (other than the portion) have been deleted, substituted, added, and / or inserted.
- the gene represented by SEQ ID NO: 59 is merely an example of a sequence including the gene encoding IL28B present in the human genome and its periphery (chromosomal antisense strand), and these mutants (deletion, This is because substitution, addition, and / or insertion) exist as individual differences.
- the P value has the same meaning as the significance probability, and is a probability value for indicating the significance of the statistical test.
- an example of a preferable marker is an oligonucleotide or a polynucleotide containing a gene polymorphism present in the polynucleotide represented by SEQ ID NO: 59.
- an oligonucleotide or polynucleotide containing a gene polymorphism present in the polynucleotide represented by SEQ ID NO: 59 was selected as an example of a preferable marker. This is because SNP numbers 42 to 55) were found in the polynucleotide represented by the sequence 59, that is, in the IL28B region and in the vicinity thereof.
- gene polymorphisms such as SNPs are often linked, it should be considered after creating a haplotype by combining not only one gene polymorphism such as one SNP but also multiple gene polymorphisms such as SNPs that are closely linked. is important. For example, by testing in combination with a gene polymorphism such as a tag (representative) SNP that expresses a haplotype in this marker (gene), the therapeutic effect of hepatitis C can be predicted more efficiently and reliably. This is because the possibility is high.
- the marker for predicting the therapeutic effect of hepatitis C containing these oligonucleotides or polynucleotides can be extracted from a patient sample and purified as necessary, but can be used in the obtained sample.
- the gene or gene fragment is mRNA, it can be used as a cDNA for reverse transcriptase and as a marker for the therapeutic effect of hepatitis C.
- the size of the oligonucleotide or polynucleotide as the marker of the present invention is preferably ⁇ 40k base length (or base pair (bp)), more preferably ⁇ 16k base length (or bp), and further preferably 1500. ⁇ 3000 bases long (or bp).
- the oligonucleotide or polynucleotide present on the human genome containing one or more gene polymorphisms selected from the above may be any oligonucleotide or polynucleotide containing at least one gene polymorphism,
- the gene polymorphism may exist in one oligonucleotide or polynucleotide, or may exist separately in a plurality of oligonucleotides or polynucleotides.
- the specific therapeutic method to be used for predicting the therapeutic effect using the marker for predicting the therapeutic effect of hepatitis C according to the present invention includes IFN monotherapy, PEGylated IFN monotherapy, PEGylated IFN / Treatment using IFN or PEGylated IFN, such as RBV (two-agent combination) therapy, PEGylated IFN / RBV / other drug (three-agent or four-agent combination) therapy, and the like.
- the marker of the present invention was found from the actual results of the two-drug combination therapy, and some of these markers are in the gene region of IL28B protein, and this IL28B is an ISG (interferon- Because it has been reported to exert an antiviral effect via stimulated genes (induction) and TLR (Toll-Like Receptor) (Ank, N. et al. J Immunol 180, 2474-2485 (2008); Marcello, T. et al. Gastroenterology 131, 1887-1898) (2006)), which is highly likely to affect the overall treatment based on IFN, and is very likely to be useful for confirming the effects of treatment methods using IFN Because it is expensive.
- ISG interferon- Because it has been reported to exert an antiviral effect via stimulated genes (induction) and TLR (Toll-Like Receptor) (Ank, N. et al. J Immunol 180, 2474-2485 (2008); Marcello, T. et al. Gastroenterology
- the possibility is high particularly in the case of SNP (SNP number 42 to 55) and other gene polymorphisms present in and around the IL28B region.
- three-drug combination therapy and four-drug combination therapy using “PEGylated IFN / RBV / one or two other drugs” are treatments based on PEGylated IFN / RBV two-drug combination therapy. In a sense, it is a kind of PEGylated IFN / RBV two-drug combination therapy.
- the marker of the present invention includes the above-mentioned IL28B-related oligo (or poly) nucleotide or other protein-encoding region
- the effect of other hepatitis C therapeutic agents other than IFN can also be determined. May be usable. This is because the proteins themselves encoded by these gene polymorphisms may be related to hepatitis C.
- oligonucleotide or polynucleotide of the present invention is an oligonucleotide or polynucleotide existing on the human genome, and includes one or more selected from the above-mentioned gene polymorphisms. , Which hybridize.
- the above-mentioned gene polymorphism means various gene polymorphisms included in the marker of the present invention.
- oligonucleotides or polynucleotides, or labels thereof have the property of hybridizing to the region containing the above-mentioned gene polymorphism, so that when predicting the therapeutic effect of hepatitis C, genes in patient samples Can be used as a probe for picking up the predictive marker of the present invention from a primer used in amplifying the gene or an amplified gene.
- a labeled substance facilitates detection of a target marker gene by binding to a probe, a primer, or the like.
- the label include biotin, digoxigenin, radioactive label, enzyme label, and fluorescent label.
- the probe of the present invention comprises the oligonucleotide or polynucleotide of the present invention or a label thereof.
- the label can be bound to the oligonucleotide or polynucleotide according to a known method.
- a probe is used to pick up a specific oligonucleotide or polynucleotide (in this case, a marker corresponding to a gene polymorphism), and is an oligonucleotide (or poly) nucleotide that is a target of detection. , Which specifically hybridizes.
- the probe sequence preferably forms a completely complementary pair with the target base sequence, but it only needs to be homologous to the complete complementary pair to the extent of hybridization.
- the probe of the present invention can be used to detect an oligo (or poly) nucleotide corresponding to a marker when predicting the therapeutic effect of hepatitis C.
- the probe can be appropriately designed so as to hybridize with the base sequence of the oligo (or poly) nucleotide (marker) to be detected.
- the size of the probe varies depending on the oligo (or poly) nucleotide to be detected, and cannot be generally specified.
- the length is ⁇ 1000 bases, preferably 5 to 500 bases, more preferably 10 to 40 bases. It may be long.
- DNA is suitable as the probe from the viewpoint of the stability of the nucleic acid molecule.
- a chimeric probe is used. It is done.
- probe Specific examples of the probe include those shown in Table 3 below when the oligo (or poly) nucleotide that is the detection target is an IL28B-related oligo (or poly) nucleotide. .
- the primer of the present invention comprises the oligonucleotide or polynucleotide of the present invention or a label thereof.
- a primer is used to amplify a specific oligonucleotide or polynucleotide (in this case, corresponding to a marker containing a gene polymorphism), and is an oligo (or poly) nucleotide that is a target for detection.
- the sense (forward) primer and the antisense (reverse) primer respectively hybridize specifically with the antisense strand and the 3 ′ end of the sense strand.
- the primer sequence is preferably one that completely forms a complementary pair with the target base sequence, but may be homologous to the complete complementary pair to the extent that it hybridizes with the target.
- the primer of the present invention can be used to amplify an oligo (or poly) nucleotide corresponding to a marker in a sample and to facilitate detection when predicting the therapeutic effect of hepatitis C.
- the primer can be appropriately designed according to the base sequence of the end of the oligo (or poly) nucleotide (marker) that is a detection target.
- Primer size The size of the primer varies depending on the oligo (or poly) nucleotide to be detected, and cannot be generally specified. For example, it is 5 to 100 bases long, preferably 10 to 70 bases long, more preferably 15 to 50 bases long. What is necessary is just about base length.
- DNA is suitable as a primer from the viewpoint of the stability of the nucleic acid molecule, but a chimeric primer is used in the isothermal gene amplification method (ICAN method).
- primers include those shown in Table 3 below when the oligo (or poly) nucleotide that is the detection target is an IL28B-related oligo (or poly) nucleotide. .
- the kit for predicting the therapeutic effect of hepatitis C according to the present invention comprises the probe and / or primer according to the present invention.
- the kit for predicting the therapeutic effect of hepatitis C is a kit used for predicting the therapeutic effect of hepatitis C.
- kits of the present invention include probes, primers, other reagents necessary for PCR (heat-resistant DNA polymerase), and devices, reagents, software, computers, etc. that are generally required for genotyping. , What you need is selected.
- the method for predicting the therapeutic effect of hepatitis C according to the present invention comprises the following steps (1) and (2).
- the “gene polymorphism present in the polynucleotide represented by SEQ ID NO: 59” included in (X) means other known publicly known genes present in the polynucleotide of SEQ ID NO: 59 other than the SNP described above.
- SNP or other genetic polymorphisms are included.
- a patient means a patient who is infected with or suspected of having hepatitis C, and includes chimpanzees, human hepatocyte-substituted chimeric mice and the like in addition to humans, but the marker of the present invention targets humans. Humans are most preferable because they are obtained by GWAS.
- Sample means blood, serum, lymph, semen, urine, saliva (including oral mucosal cells), other body fluids, and biological tissues, but blood (lymphocytes) is the easiest to collect and is invasive Is preferable in that it is low.
- Oligo (or poly) nucleotides are prepared from the sample.
- examples of the oligo (or poly) nucleotide include genomic DNA or cDNA prepared by reverse transcriptase based on mRNA.
- Preparation of oligo (or poly) nucleotides may be carried out according to a known method, but when the oligo (or poly) nucleotide is DNA, for example, from blood cells such as lymphocytes and granulocytes in the blood sample. , Salting out, PCI (phenol chloroform extraction) method, a method using a commercially available DNA extraction kit, etc., and other known methods.
- DNA for example, from blood cells such as lymphocytes and granulocytes in the blood sample.
- PCI phenol chloroform extraction
- oligo (or poly) nucleotide is mRNA
- the method of acidifying the solution in the PCI method and extracting from the aqueous layer the method using an oligo dT column, the method using a commercially available RNA extraction kit, etc.
- Known methods can be used.
- nucleic acid obtained from the sample is small, it can be amplified by a known method, if necessary.
- a PCR method or the like can be used.
- the RT-PCR method can be used.
- the quantitative PCR method a real-time PCR method using TaqMan probe or SYBR Green (Power SYBR) can also be used.
- a known sequencing method or the like can be used.
- a direct sequencing method in which a base sequence is directly determined using a sequencer or the like, RELP (restriction fragment length polymorphism) PCR-RELP method, TaqMan method, DigiTag2 method, single nucleotide primer extension method, SnaPshot method (ABI), ASP-PCR method, PCR-SSO (sequence specific oligonucleotide) method, PCR-SSP (specific sequence primer) ) Method, PCR-SSCP (single-stranded conformational polymorphism analysis) method, confirmation of nucleic acid length by electrophoresis, etc., and method of using genetic polymorphism testing equipment such as DNA chip, etc. It is not limited.
- Therapeutic method to be used for predicting the therapeutic effect by the method of the present invention includes IFN monotherapy, PEGylated IFN monotherapy, PEGylated IFN / RBV (two-drug combination) ) Treatment, treatment using IFN or PEGylated IFN, such as therapy, PEGylated IFN / RBV / other drugs (3 drugs or 4 drugs combined) therapy, and the like.
- the marker of the present invention contains the above-mentioned IL28B-related oligo (or poly) nucleotide or other protein-encoding region, it is also used for determining the effect of other hepatitis C therapeutic agents other than IFN. There is a possibility. This is because the proteins themselves encoded by these gene polymorphisms may be related to hepatitis C.
- the “gene for searching for marker for predicting therapeutic effect of hepatitis C” of the present invention is characterized by having the following sequence (A) or (B).
- Oligonucleotide or polynucleotide (B) (A) comprising at least a part of the polynucleotide represented by SEQ ID NO: 59, wherein one to several nucleotides are deleted, substituted, added, and / or Inserted oligonucleotide or polynucleotide
- the gene represented by SEQ ID NO: 59 is a sequence (IL28B-related oligo (or poly) nucleotide) containing the gene encoding L28B and its periphery.
- “Screening method for therapeutic agent for hepatitis C of the present invention” The method for screening for a therapeutic agent for hepatitis C of the present invention is characterized by using at least one of the following (A) to (C).
- A Oligonucleotide or polynucleotide
- B (A) comprising at least a part of the polynucleotide represented by SEQ ID NO: 59, wherein one to several nucleotides are deleted, substituted, added, and / or Inserted oligonucleotide or polynucleotide
- C oligonucleotide or polynucleotide present on the human genome, including rs8103142
- IL28B protein encoded by the gene in which SRS rs8103142 exists
- oligo (or poly) nucleotides containing rs8103142 such as IL28B This is because there is a possibility that it can also be used to determine the effect of the therapeutic agent for hepatitis C.
- a screening method various known methods for selecting a substance that increases or decreases the expression level of a gene or protein and the activity of the protein can be used. “In vivo”, “ex vivo”, “cell-free-” Any experimental system such as “system” may be used.
- the preventive or therapeutic agent for hepatitis C of the present invention is characterized by containing IL28B protein and / or a gene encoding the same.
- IL28 is significantly higher than in patients having a major allele homozygote (high therapeutic effect group).
- the expression level of IL28B is likely to be significantly reduced, and as described above, it is highly likely that IL28B itself is related to hepatitis C. From this, it is considered that administration of IL28B or a gene encoding it is likely to be useful for the treatment of hepatitis C.
- SNP Array 6.0 Affymetrix Genome-Wide Human SNP Array 6.0 (hereinafter referred to as SNP Array 6.0) equipped with more than 900,000 SNP analysis probes SNP typing of 154 people, classified into subgroups (GWAS Stage: 1st panel: 82 ineffective (NVR) group vs. 72 in effective (VR) group) according to drug responsiveness, genome-wide association analysis (GWAS )
- Table 1 shows the results of selecting a P value of 10 -5 level or lower.
- the P value was calculated mainly by chi-square test.
- the ineffective group means an example in which the virus has never disappeared during treatment.
- QC call rate means the percentage of SNPs that can be analyzed.
- the SNP genotype was determined using the DigiTag2 method for the purpose of validation and replication of GWAS data.
- the DigiTag2 method is a multiplex SNP typing method that can determine the genotype of multiple SNPs at once using SNP-specific common probe and allele-specific query probe for each SNP to be analyzed. (Analytical Biochemistry vol.364, No.1, 2007, P.78-85, etc.).
- the detection probe is divided into a 3′-side probe (common probe) existing downstream and adjacent to the SNP position, and an upstream 5 ′ probe (query probe) containing SNP at the 3 ′ end.
- query probe corresponding to alleles Two types are prepared for each SNP. Depending on the SNP genotype corresponding to the 3 ′ end of query probe, only query probe with a complementary base can bind to common probe that is adjacent on the 3 ′ side.
- the query probe designed for the antisense strand has a base sequence that is homologous to the Fasta sequence in the NCBI database. When designed for the sense strand, it becomes a complementary base sequence.
- two types of query tags are linked to the 5 'end of each query tag, corresponding to alleles, and a different common tag for each SNP is linked to the 3' end of common tag. Yes.
- the binding product of query probe and common probe generated according to the genotype of the specimen is captured by oligo DNA having a base sequence complementary to common tag on the DNA chip.
- a is for 184 healthy Japanese people quoted from Nishida, N. et al. Evaluating the performance of Affymetrix SNP Array 6.0 platform with 400 Japanese individuals. BMC Genomics 9, 431 (2008). Means the frequency of minor alleles of each SNP (MAF: Minor Allele Frequency).
- C in Table 2 means a P value calculated by Pearson's chi-square test in the dominant effect model.
- SNP type in Table 3 means the following.
- gSNP Genomic SNP, SNP in a region other than the expressed gene region
- iSNP intron SNP, SNP in the intron region
- nsSNP nonsynonymous SNP, SNP in the translation region that causes amino acid substitution
- rSNP regulatory SNP, SNP in the promoter region of the expressed gene
- primers and probes of the present invention are not limited to these, and 1 to It goes without saying that the primers and probes of the present invention also include those in which several nucleotides have been deleted, substituted, added, and / or inserted and can hybridize with the markers of the present invention. There is no.
- the primers and probes in Table 3 are DNA as shown in the sequence listing, but may be RNA having the same base, or a chimera of DNA and RNA. However, DNA is preferable from the viewpoint of stability and the like.
- the marker of the present invention is said to have a considerably small P value and a high predictive predictive value compared with the conventional marker for predicting therapeutic effect of hepatitis C. be able to.
- 11 SNPs with SNP numbers 42 to 52 shown in Table 3 have particularly small P values
- 7 SNPs with SNPs 45 to 51 have extremely small P values, and these have extremely high predictive predictive values. is there.
- IL28B or its gene is highly likely to be useful as a preventive or therapeutic agent for hepatitis C.
- the present invention is capable of predicting the therapeutic effect of hepatitis C with higher accuracy, particularly in “IFN therapy”, particularly “PEGylated IFN / RBV combination therapy”, which is currently the mainstream.
- IFN therapy particularly “PEGylated IFN / RBV combination therapy”
- the clinical significance is great because it enables an unprecedented accurate prediction.
- Sequence number 1 sense primer for SNP42: 5 'TATAAAGAAAAGGACAGGCCACTTTGGTACGCCAA 3' Sequence number 2: antisense primer for SNP42: 5 'TAACCTCTCTGAGTACCAGCTTCTTCATTTTCAAGGTAGAA 3' Sequence number 3: sense primer for SNP43: 5 'TGGGTTGTCCACCTGGACCCAATGTCATCACAAAA 3' Sequence number 4: antisense primer for SNP43: 5 'GGAGGGAAGGAAGTTCTGACACATGCTACAATAAAAATGAA 3' Sequence number 5: sense primer for SNP44,45,46: 5 'GTCAGCCATCCTCCAATCCCATCAGAGTGGTCTAA 3' Sequence number 6: antisense primer for SNP44,45,46: 5 'TCTCAGGTTGCATGACTGGCGGAAGGGTCAGACACA 3' Sequence number
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Organic Chemistry (AREA)
- General Health & Medical Sciences (AREA)
- Engineering & Computer Science (AREA)
- Medicinal Chemistry (AREA)
- Zoology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Pharmacology & Pharmacy (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Genetics & Genomics (AREA)
- Animal Behavior & Ethology (AREA)
- Wood Science & Technology (AREA)
- Public Health (AREA)
- Veterinary Medicine (AREA)
- Immunology (AREA)
- Virology (AREA)
- Molecular Biology (AREA)
- Gastroenterology & Hepatology (AREA)
- Biochemistry (AREA)
- General Chemical & Material Sciences (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Epidemiology (AREA)
- Nuclear Medicine, Radiotherapy & Molecular Imaging (AREA)
- Biotechnology (AREA)
- Analytical Chemistry (AREA)
- Microbiology (AREA)
- General Engineering & Computer Science (AREA)
- Biomedical Technology (AREA)
- Communicable Diseases (AREA)
- Pathology (AREA)
- Oncology (AREA)
- Physics & Mathematics (AREA)
- Biophysics (AREA)
- Measuring Or Testing Involving Enzymes Or Micro-Organisms (AREA)
- Medicines That Contain Protein Lipid Enzymes And Other Medicines (AREA)
- Pharmaceuticals Containing Other Organic And Inorganic Compounds (AREA)
Abstract
より精度の高い治療効果予測マーカー、及びそれを用いた、「PEG化IFN/RBV併用療法」における、より精度の高いC型肝炎治療効果の予測方法等を提供する。ヒトゲノム上に存在するオリゴヌクレオチド又はポリヌクレオチドであって、下記に示す遺伝子多型,又はこれらと連鎖不平衡状態(R2>0.8)にあるものから選択される1種又は2種以上を含むものであることを特徴とする、C型肝炎の治療効果を予測するためのマーカー,及びそれを用いたC型肝炎の治療効果予測方法。rs12043519, rs1986953, rs13392065, rs16987149, rs16987155, rs2374341, rs2374342, rs7566701, rs12713624, rs10208788, rs10208883, rs9876122, rs2036081, rs13122167, rs4488950, rs13150115, rs7663113, rs1379606, rs913500, rs6458003, rs6906840, rs2224068, rs9390164, rs4512312, rs757844, rs4876271, rs7942675, rs17787293, rs917334, rs6489019, rs6489020, rs6489021, rs11610391, rs9528038, rs341554, rs2325219, rs1353348, rs2076954, rs12907066, rs8075713, rs4795794, rs955155, rs12972991, rs12980275, rs8105790, rs11881222, rs8103142, rs28416813, rs4803219, rs8099917, rs7248668, rs10853728, rs4803223, rs12980602, rs4803224, 配列番号59で表されるポリヌクレオチド内に存在する遺伝子多型。
Description
本発明は、宿主ゲノム全体に亘って網羅的に検証した、C型肝炎の治療効果を予測するためのマーカー,及びそれを用いた、より精度の高い、C型肝炎の治療効果の予測を行う方法,並びに当該予測に用いるプローブ,プライマー,もしくはそれらを含むキット,並びにC型肝炎の予防又は治療剤等に関するものである。
本出願は、参照によりここに援用されるところの日本出願特願2009-192615号優先権を請求する。
C型肝炎に対する治療効果を予測する方法は、従来、種々検討されてきたが、その多くは、患者が罹患した“ウイルス側因子”をもとに判断するものが主流であった。
具体的には、ウイルス遺伝子型や特定の領域(コア領域や非構造領域(NS5A))の変異によりインターフェロン(IFN)の治療効果を予測してきた。
具体的には、ウイルス遺伝子型や特定の領域(コア領域や非構造領域(NS5A))の変異によりインターフェロン(IFN)の治療効果を予測してきた。
近年、“患者(宿主)自身の一塩基多型(SNP)”を利用する方法が報告されているが、いずれも「IFN単独療法」における治療効果を予測するものであって、現在主流となっている、「PEG化IFN/RBV併用療法」を基準としたものでは無く、そのまま転用することは不可能であった(特許文献1,非特許文献1,2等)。
「IFN単独療法」と「PEG化IFN/RBV併用療法」では、SNPの判断基準となる治癒効率(著効率)自体が全く異なるため、「IFN単独療法」に対して導きだされたSNPを「PEG化IFN/RBV併用療法」を対象とするSNPに転用することはできないからである。
「IFN単独療法」と「PEG化IFN/RBV併用療法」では、SNPの判断基準となる治癒効率(著効率)自体が全く異なるため、「IFN単独療法」に対して導きだされたSNPを「PEG化IFN/RBV併用療法」を対象とするSNPに転用することはできないからである。
また、これら従来の方法は、特定の遺伝子を候補として検討したに過ぎず、宿主ゲノム全体に亘る網羅的な検証では無かったため、精度の点で問題が残っていた。
薬物による治療効果には、複数の要因が関連していることが多く、ゲノム全体に亘る網羅的なSNP検証をしないと、正確な治療効果の予測とは言えないからである。
薬物による治療効果には、複数の要因が関連していることが多く、ゲノム全体に亘る網羅的なSNP検証をしないと、正確な治療効果の予測とは言えないからである。
Matsuyama, N. et al. Hepatol Res 25, 221-225, 2003
Tsukada, H. et al. Gastroenterology 136, 1796-1805, 2009
本発明者等は、上記問題を解決すべく、日本人を中心とするアジア系集団において、「PEG化IFN/RBV併用療法」についてのSNP解析を、宿主ゲノム全体に亘って網羅的に実施(Genome-Wide Association Study:GWAS)した結果、危険率の高い(治療効果が得られ難い)SNPを多数発見するとともに、当該SNPの集中している遺伝子領域を見出し、本発明に到達したものであって、その目的とするところは、SNPやその他の遺伝子多型を利用した、より精度の高いC型肝炎治療効果予測用マーカー,当該予測に用いるプローブ,プライマー,又はキット,当該マーカーを用いたC型肝炎治療効果の予測方法,及びその治療効果予測マーカーの探索用遺伝子,更には本発明のSNPやその他の遺伝子多型の集中する領域がコードするタンパク質を用いたC型肝炎の予防又は治療剤を提供するにある。
上記の目的は、下記第一の発明乃至第十四の発明によって達成される。
<第一の発明>
ヒトゲノム上に存在するオリゴヌクレオチド又はポリヌクレオチドであって、下記に示す遺伝子多型,又はこれらと連鎖不平衡状態(R2>0.8)にあるものから選択される1種又は2種以上を含むものであることを特徴とする、C型肝炎の治療効果を予測するためのマーカー。
rs12043519, rs1986953, rs13392065, rs16987149, rs16987155, rs2374341, rs2374342, rs7566701, rs12713624, rs10208788, rs10208883, rs9876122, rs2036081, rs13122167, rs4488950, rs13150115, rs7663113, rs1379606, rs913500, rs6458003, rs6906840, rs2224068, rs9390164, rs4512312, rs757844, rs4876271, rs7942675, rs17787293, rs917334, rs6489019, rs6489020, rs6489021, rs11610391, rs9528038, rs341554, rs2325219, rs1353348, rs2076954, rs12907066, rs8075713, rs4795794, rs955155, rs12972991, rs12980275, rs8105790, rs11881222, rs8103142, rs28416813, rs4803219, rs8099917, rs7248668, rs10853728, rs4803223, rs12980602,rs4803224,配列番号59で表されるポリヌクレオチド内に存在する遺伝子多型
ヒトゲノム上に存在するオリゴヌクレオチド又はポリヌクレオチドであって、下記に示す遺伝子多型,又はこれらと連鎖不平衡状態(R2>0.8)にあるものから選択される1種又は2種以上を含むものであることを特徴とする、C型肝炎の治療効果を予測するためのマーカー。
rs12043519, rs1986953, rs13392065, rs16987149, rs16987155, rs2374341, rs2374342, rs7566701, rs12713624, rs10208788, rs10208883, rs9876122, rs2036081, rs13122167, rs4488950, rs13150115, rs7663113, rs1379606, rs913500, rs6458003, rs6906840, rs2224068, rs9390164, rs4512312, rs757844, rs4876271, rs7942675, rs17787293, rs917334, rs6489019, rs6489020, rs6489021, rs11610391, rs9528038, rs341554, rs2325219, rs1353348, rs2076954, rs12907066, rs8075713, rs4795794, rs955155, rs12972991, rs12980275, rs8105790, rs11881222, rs8103142, rs28416813, rs4803219, rs8099917, rs7248668, rs10853728, rs4803223, rs12980602,rs4803224,配列番号59で表されるポリヌクレオチド内に存在する遺伝子多型
<第二の発明>
オリゴヌクレオチド又はポリヌクレオチドの長さが、40k塩基長以下であることを特徴とする、第一の発明に記載の、C型肝炎の治療効果を予測するためのマーカー。
オリゴヌクレオチド又はポリヌクレオチドの長さが、40k塩基長以下であることを特徴とする、第一の発明に記載の、C型肝炎の治療効果を予測するためのマーカー。
<第三の発明>
C型肝炎の治療効果が、インターフェロン及び/又はPEG化インターフェロンを用いた治療による治療効果であることを特徴とする、第一の発明又は第二の発明に記載の、C型肝炎の治療効果を予測するためのマーカー。
C型肝炎の治療効果が、インターフェロン及び/又はPEG化インターフェロンを用いた治療による治療効果であることを特徴とする、第一の発明又は第二の発明に記載の、C型肝炎の治療効果を予測するためのマーカー。
<第四の発明>
C型肝炎の治療効果が、PEG化インターフェロン/RBV併用療法による治療効果であることを特徴とする、第一の発明乃至第三の発明のいずれかに記載の、C型肝炎の治療効果を予測するためのマーカー。
C型肝炎の治療効果が、PEG化インターフェロン/RBV併用療法による治療効果であることを特徴とする、第一の発明乃至第三の発明のいずれかに記載の、C型肝炎の治療効果を予測するためのマーカー。
<第五の発明>
ヒトゲノム上に存在するオリゴヌクレオチド又はポリヌクレオチドであって第一の発明に記載の遺伝子多型から選択される1種又は2種以上を含むものに、ハイブリダイズするオリゴヌクレオチド若しくはポリヌクレオチド,又はその標識物。
ヒトゲノム上に存在するオリゴヌクレオチド又はポリヌクレオチドであって第一の発明に記載の遺伝子多型から選択される1種又は2種以上を含むものに、ハイブリダイズするオリゴヌクレオチド若しくはポリヌクレオチド,又はその標識物。
<第六の発明>
第五の発明に記載のオリゴヌクレオチド若しくはポリヌクレオチド,又はその標識物からなるプローブ。
第五の発明に記載のオリゴヌクレオチド若しくはポリヌクレオチド,又はその標識物からなるプローブ。
<第七の発明>
第五の発明に記載のオリゴヌクレオチド若しくはポリヌクレオチド,又はその標識物からなるプライマー。
第五の発明に記載のオリゴヌクレオチド若しくはポリヌクレオチド,又はその標識物からなるプライマー。
<第八の発明>
第六の発明に記載のプローブ及び/又は第七の発明に記載のプライマーを含むことを特徴とする、C型肝炎の治療効果予測用キット。
第六の発明に記載のプローブ及び/又は第七の発明に記載のプライマーを含むことを特徴とする、C型肝炎の治療効果予測用キット。
<第九の発明>
下記(1)及び(2)の工程を有することを特徴とする、C型肝炎の治療効果の予測を行う方法。
(1)患者から得たサンプルにより、下記(X)の遺伝子型を決定する工程。
(X)第一の発明に記載の遺伝子多型の少なくとも1種
(2)(1)で決定された遺伝子型から、C型肝炎の治療効果の予測を行う工程。
下記(1)及び(2)の工程を有することを特徴とする、C型肝炎の治療効果の予測を行う方法。
(1)患者から得たサンプルにより、下記(X)の遺伝子型を決定する工程。
(X)第一の発明に記載の遺伝子多型の少なくとも1種
(2)(1)で決定された遺伝子型から、C型肝炎の治療効果の予測を行う工程。
<第十の発明>
C型肝炎の治療効果が、インターフェロン及び/又はPEG化インターフェロンを用いた治療による治療効果であることを特徴とする、第九の発明に記載の、C型肝炎の治療効果の予測を行う方法。
C型肝炎の治療効果が、インターフェロン及び/又はPEG化インターフェロンを用いた治療による治療効果であることを特徴とする、第九の発明に記載の、C型肝炎の治療効果の予測を行う方法。
<第十一の発明>
C型肝炎の治療効果が、PEG化インターフェロン/RBV併用療法による治療効果であることを特徴とする、第十の発明に記載の、C型肝炎の治療効果の予測を行う方法。
C型肝炎の治療効果が、PEG化インターフェロン/RBV併用療法による治療効果であることを特徴とする、第十の発明に記載の、C型肝炎の治療効果の予測を行う方法。
<第十二の発明>
下記の(A)又は(B)の配列を有することを特徴とする、C型肝炎の治療効果予測マーカーの探索用遺伝子。
(A)配列番号59で表されるポリヌクレオチドの少なくとも一部を含むオリゴヌクレオチド又はポリヌクレオチド
(B)(A)のうち、1乃至数個のヌクレオチドが、欠失,置換,付加,及び/又は挿入された、オリゴヌクレオチド又はポリヌクレオチド
下記の(A)又は(B)の配列を有することを特徴とする、C型肝炎の治療効果予測マーカーの探索用遺伝子。
(A)配列番号59で表されるポリヌクレオチドの少なくとも一部を含むオリゴヌクレオチド又はポリヌクレオチド
(B)(A)のうち、1乃至数個のヌクレオチドが、欠失,置換,付加,及び/又は挿入された、オリゴヌクレオチド又はポリヌクレオチド
<第十三の発明>
下記(A)乃至(C)の少なくとも1種を用いることを特徴とする、C型肝炎治療薬のスクリーニング方法。
(A)配列番号59で表されるポリヌクレオチドの少なくとも一部を含むオリゴヌクレオチド又はポリヌクレオチド
(B)(A)のうち、1乃至数個のヌクレオチドが、欠失,置換,付加,及び/又は挿入された、オリゴヌクレオチド又はポリヌクレオチド
(C)ヒトゲノム上に存在するオリゴヌクレオチド又はポリヌクレオチドであってrs8103142を含むもの
下記(A)乃至(C)の少なくとも1種を用いることを特徴とする、C型肝炎治療薬のスクリーニング方法。
(A)配列番号59で表されるポリヌクレオチドの少なくとも一部を含むオリゴヌクレオチド又はポリヌクレオチド
(B)(A)のうち、1乃至数個のヌクレオチドが、欠失,置換,付加,及び/又は挿入された、オリゴヌクレオチド又はポリヌクレオチド
(C)ヒトゲノム上に存在するオリゴヌクレオチド又はポリヌクレオチドであってrs8103142を含むもの
<第十四の発明>
IL28Bタンパク質及び/又はそれをコードする遺伝子を含むことを特徴とする、C型肝炎の予防又は治療剤。
IL28Bタンパク質及び/又はそれをコードする遺伝子を含むことを特徴とする、C型肝炎の予防又は治療剤。
本発明の「C型肝炎の治療効果を予測するためのマーカー」は、宿主ゲノム全体に亘って網羅的に検証した結果に基づくため、C型肝炎の治療,特にインターフェロン療法,中でも、最新治療である「PEG化IFN/RBV併用療法」の治療効果をより正確に予測できる指標となる。
また、危険度の値が、従来のマーカーより明確で(P値がかなり小さい)、判断が容易である。
本発明の「プローブ」,「プライマー」,それらを用いた「C型肝炎の治療効果予測用キット」,及び「C型肝炎の治療効果の予測を行う方法」によって、C型肝炎の治療,特にインターフェロン療法,中でも、最新治療である「PEG化IFN/RBV併用療法」の治療効果をより正確に予測できる。
本発明の「C型肝炎の治療効果予測マーカーの探索用遺伝子」内を、更に綿密に調査することによって、より迅速に、C型肝炎治療効果予測用の、新たなマーカーを発見し得る。
本発明の「C型肝炎治療薬のスクリーニング方法」によって、インターフェロンやRBV以外の、C型肝炎治療薬のスクリーニングも可能となる。
本発明の予防又は治療剤によって、C型肝炎の効果的な予防又は治療が期待できる。
また、危険度の値が、従来のマーカーより明確で(P値がかなり小さい)、判断が容易である。
本発明の「プローブ」,「プライマー」,それらを用いた「C型肝炎の治療効果予測用キット」,及び「C型肝炎の治療効果の予測を行う方法」によって、C型肝炎の治療,特にインターフェロン療法,中でも、最新治療である「PEG化IFN/RBV併用療法」の治療効果をより正確に予測できる。
本発明の「C型肝炎の治療効果予測マーカーの探索用遺伝子」内を、更に綿密に調査することによって、より迅速に、C型肝炎治療効果予測用の、新たなマーカーを発見し得る。
本発明の「C型肝炎治療薬のスクリーニング方法」によって、インターフェロンやRBV以外の、C型肝炎治療薬のスクリーニングも可能となる。
本発明の予防又は治療剤によって、C型肝炎の効果的な予防又は治療が期待できる。
以下、本発明について詳細に説明する。
尚、本発明において、「遺伝子」,「オリゴヌクレオチド」,及び「ポリヌクレオチド」とは、アデニン(A),グアニン(G)等のプリン塩基や、チミン(T),ウラシル(U),シトシン(C)等のピリミジン塩基やそれらの修飾塩基を構成要素として含むものであり、一本鎖又は二本鎖のDNA,一本鎖又は二本鎖のRNA,一本鎖DNAと一本鎖RNAからなるハイブリッド体,RNAとDNAが結合して一本鎖となったキメラ体のいずれか、又は複数を意味するものである。また、DNAには、cDNAも含まれる。
尚、本発明において、「遺伝子」,「オリゴヌクレオチド」,及び「ポリヌクレオチド」とは、アデニン(A),グアニン(G)等のプリン塩基や、チミン(T),ウラシル(U),シトシン(C)等のピリミジン塩基やそれらの修飾塩基を構成要素として含むものであり、一本鎖又は二本鎖のDNA,一本鎖又は二本鎖のRNA,一本鎖DNAと一本鎖RNAからなるハイブリッド体,RNAとDNAが結合して一本鎖となったキメラ体のいずれか、又は複数を意味するものである。また、DNAには、cDNAも含まれる。
[本発明のC型肝炎の治療効果を予測するためのマーカー]
《マーカーの定義》
本発明の「C型肝炎の治療効果を予測するためのマーカー」は、C型肝炎の治療効果を予測するための指標となるものであって、ヒトゲノム上に存在するオリゴヌクレオチド又はポリヌクレオチドであって、下記に示す遺伝子多型,又はこれらと連鎖不平衡状態(R2>0.8)にあるものから選択される1種又は2種以上を含むものである。
《マーカーの定義》
本発明の「C型肝炎の治療効果を予測するためのマーカー」は、C型肝炎の治療効果を予測するための指標となるものであって、ヒトゲノム上に存在するオリゴヌクレオチド又はポリヌクレオチドであって、下記に示す遺伝子多型,又はこれらと連鎖不平衡状態(R2>0.8)にあるものから選択される1種又は2種以上を含むものである。
尚、特定の遺伝子多型と「連鎖不平衡状態(R2>0.8)にあるもの」は、当該特定の遺伝子多型と同様の治療効果予測が可能なものであるが、例えばHaploviewソフトウェア等によって、公知の方法に従って検出できる。
具体的には、マーカーとは、C型肝炎の感染者又は感染が疑われる対象者の、血液等のサンプル中に存在している遺伝子又は遺伝子断片であって、後述するプローブ等によって検出され、その遺伝子型(SNP等の遺伝子多型)を調べることによってC型肝炎の治療効果を予測するための試料となるものである。
rs12043519, rs1986953, rs13392065, rs16987149, rs16987155, rs2374341, rs2374342, rs7566701, rs12713624, rs10208788, rs10208883, rs9876122, rs2036081, rs13122167, rs4488950, rs13150115, rs7663113, rs1379606, rs913500, rs6458003, rs6906840, rs2224068, rs9390164, rs4512312, rs757844, rs4876271, rs7942675, rs17787293, rs917334, rs6489019, rs6489020, rs6489021, rs11610391, rs9528038, rs341554, rs2325219, rs1353348, rs2076954, rs12907066, rs8075713, rs4795794, rs955155, rs12972991, rs12980275, rs8105790, rs11881222, rs8103142, rs28416813, rs4803219, rs8099917, rs7248668, rs10853728, rs4803223, rs12980602,rs4803224:(SNP番号1~55:表1,3参照),配列番号59で表されるポリヌクレオチド内に存在する遺伝子多型
上記のrs番号で示されている遺伝子多型は、SNPを表す。
また、配列番号59とは、ヒトゲノムに存在するIL28Bをコードする遺伝子及びその周辺を含む配列の一例である。
また、配列番号59とは、ヒトゲノムに存在するIL28Bをコードする遺伝子及びその周辺を含む配列の一例である。
尚、IL28Bは、2本の染色体中、いわゆるアンチセンス鎖に載っている遺伝子である。
一方、表3においては、アリルは、染色体のセンス鎖で表現している。
従って、配列番号59内に存在する4つのSNP(rs11881222, rs8103142, rs28416813, rs4803219)のメジャーアリルの記載(図2,3の枠で囲った部位)は、表3で示したメジャーアリルと、相補的になっている。
一方、表3においては、アリルは、染色体のセンス鎖で表現している。
従って、配列番号59内に存在する4つのSNP(rs11881222, rs8103142, rs28416813, rs4803219)のメジャーアリルの記載(図2,3の枠で囲った部位)は、表3で示したメジャーアリルと、相補的になっている。
上記の「遺伝子多型」とは、普通とは違った遺伝子型であって、その頻度が母集団の1%以上であるものを意味し、SNPのみならず、1乃至数個のヌクレオチドの欠失,置換,付加,及び/又は挿入等の変異の他、VNTR(variable number of tandem repeat:反復配列多型)やSTRP(short tandem repeat polymorphism:マイクロサテライト多型)等をも含むものであり、反復多型部分の一部が、欠失,置換,付加,及び/又は挿入等の変異を受けているものも含む。
このような、SNP以外の遺伝子多型をも本発明のマーカーに含めたのは、配列番号59で表されるポリヌクレオチドのプロモーター領域に、数個のヌクレオチドの欠失が存在する場合があり、この欠失は、上述のSNPのうちの一部のSNPのリスクアリルとの連鎖が見られ、C型肝炎治療への抵抗性に関連する遺伝子多型である可能性が高いと考えられるからである。
具体的には、配列番号59内に存在する、IL28B遺伝子の、開始コドン上流のプロモーター領域に存在する「TATAの繰り返し配列(-595~-568位)」(図2の、枠で囲った300番目~327番目の配列)中に、4個の欠失が見られる場合があり、しかも、この欠失は、上述のSNPのうちP値のかなり低い、rs11881222, rs8103142, rs28416813, rs4803219のマイナーアリル(リスクアリル)との連鎖が見られた。
具体的には、配列番号59内に存在する、IL28B遺伝子の、開始コドン上流のプロモーター領域に存在する「TATAの繰り返し配列(-595~-568位)」(図2の、枠で囲った300番目~327番目の配列)中に、4個の欠失が見られる場合があり、しかも、この欠失は、上述のSNPのうちP値のかなり低い、rs11881222, rs8103142, rs28416813, rs4803219のマイナーアリル(リスクアリル)との連鎖が見られた。
尚、「配列番号59で表されるポリヌクレオチド内に存在する遺伝子多型」とは、「配列番号59で表されるポリヌクレオチド内に存在する各種の遺伝子多型のうち、上記で列挙したSNPを除くもの」であり、他の公知の又は未知の、SNPその他の遺伝子多型を意味する。
このような遺伝子多型を含めたのは、上記したSNPの多くが配列番号59のポリヌクレオチド領域内に存在していることから、配列番号59自体がC型肝炎の治療効果に関わっている可能性が高く、この領域内の遺伝子多型であれば、C型肝炎の治療効果予測に関与している可能性が高いと考えられるからである。
また、「配列番号59で表されるポリヌクレオチド内に存在する遺伝子多型」を含む本発明のマーカーには、当然ながら、配列番号59で表されるポリヌクレオチドのうち、(遺伝子多型を示す部分以外の)1乃至数個のヌクレオチドが、欠失,置換,付加,及び/又は挿入された、オリゴヌクレオチド又はポリヌクレオチドも含まれる。
配列番号59で表される遺伝子は、あくまでもヒトゲノムに存在するIL28Bをコードする遺伝子及びその周辺を含む配列の一例(染色体のアンチセンス鎖)であり、ヒトゲノム上では、これらの変異体(欠失,置換,付加,及び/又は挿入)が個人差として存在するからである。
尚、上記SNPを表す数値(rs番号)や、配列表に記載の配列は、NCBIのデータベース(Build 35)に掲載されているヒトの遺伝子情報を参酌したものである。
上記の中でも、
rs955155, rs12972991, rs12980275, rs8105790, rs11881222, rs8103142, rs28416813, rs4803219, rs8099917, rs7248668, rs10853728:(SNP番号42~52:表3参照)
の11SNPsが、治療効果の重要な指標であるいわゆるP値(P value)が小さく(=「-log P」が大)、C型肝炎の治療効果に差が出やすいと考えられるため、マーカーとしては好ましく、中でも
rs8105790, rs11881222, rs8103142, rs28416813, rs4803219, rs8099917, rs7248668 :(SNP番号45~51:表3参照)
の7SNPsは、ゲノムワイド関連解析の有意水準(Bonferroni criterion P <8.05 ×10-8 (0.05/621,220))を満たし、かつ同じハプロブロック内に存在することより、重要なマーカーとして特に好ましい。
rs955155, rs12972991, rs12980275, rs8105790, rs11881222, rs8103142, rs28416813, rs4803219, rs8099917, rs7248668, rs10853728:(SNP番号42~52:表3参照)
の11SNPsが、治療効果の重要な指標であるいわゆるP値(P value)が小さく(=「-log P」が大)、C型肝炎の治療効果に差が出やすいと考えられるため、マーカーとしては好ましく、中でも
rs8105790, rs11881222, rs8103142, rs28416813, rs4803219, rs8099917, rs7248668 :(SNP番号45~51:表3参照)
の7SNPsは、ゲノムワイド関連解析の有意水準(Bonferroni criterion P <8.05 ×10-8 (0.05/621,220))を満たし、かつ同じハプロブロック内に存在することより、重要なマーカーとして特に好ましい。
P値とは、有意確率と同一の意味であり、統計学的検定の有意性を示すための確率値である。
この他、配列番号59で表されるポリヌクレオチド内に存在する遺伝子多型を含むオリゴヌクレオチド又はポリヌクレオチドが、好ましいマーカーの一例として挙げられる。
配列番号59で表されるポリヌクレオチド内に存在する遺伝子多型を含むオリゴヌクレオチド又はポリヌクレオチドを、好ましいマーカーの一例として選んだのは、上述した通り、SNP番号1~55のSNPのうち多数(SNP番号42~55)が、配列59で表されるポリヌクレオチド,つまりIL28B領域内及びその近傍で見つかったからである。
SNP等の遺伝子多型は連鎖することが多いため、1つのSNP等の遺伝子多型だけでなく近接し連鎖する複数のSNP等の遺伝子多型を組み合わせ、ハプロタイプを作った上で、検討することが重要である。例えば、このマーカー(遺伝子)内のハプロタイプを表現するタグ(代表)SNP等の遺伝子多型を組み合わせて検査することで、C型肝炎の治療効果の予測が、より一層、効率的かつ確実となる可能性が高いからである。
これらのオリゴヌクレオチド又はポリヌクレオチドを含むC型肝炎の治療効果予測マーカーは、患者サンプルから抽出及び必要に応じて精製して得られるオリゴヌクレオチド又はポリヌクレオチドを利用できるが、得られたサンプル中に存在している遺伝子又は遺伝子断片がmRNAである場合には、逆転写酵素でcDNAとして、C型肝炎の治療効果のマーカーとして用いることができる。
《マーカーの大きさ》
上記の本発明のマーカーとしてのオリゴヌクレオチド又はポリヌクレオチドの大きさは、~40k塩基長(又は塩基対(bp))が好ましく、より好ましくは、~16k塩基長(又はbp),更に好ましくは1500~3000塩基長(又はbp)である。
上記の本発明のマーカーとしてのオリゴヌクレオチド又はポリヌクレオチドの大きさは、~40k塩基長(又は塩基対(bp))が好ましく、より好ましくは、~16k塩基長(又はbp),更に好ましくは1500~3000塩基長(又はbp)である。
《マーカーの種類》
上記から選択される1種又は2種以上の遺伝子多型を含む、ヒトゲノム上に存在するオリゴヌクレオチド又はポリヌクレオチドは、遺伝子多型を少なくとも1種含むオリゴヌクレオチド又はポリヌクレオチドであれば良く、複数の遺伝子多型が、1つのオリゴヌクレオチド又はポリヌクレオチド中に存在していても、複数のオリゴヌクレオチド又はポリヌクレオチド中に分かれて存在していても良い。
上記から選択される1種又は2種以上の遺伝子多型を含む、ヒトゲノム上に存在するオリゴヌクレオチド又はポリヌクレオチドは、遺伝子多型を少なくとも1種含むオリゴヌクレオチド又はポリヌクレオチドであれば良く、複数の遺伝子多型が、1つのオリゴヌクレオチド又はポリヌクレオチド中に存在していても、複数のオリゴヌクレオチド又はポリヌクレオチド中に分かれて存在していても良い。
《マーカーによって治療効果を予測する対象となる治療方法》
本発明のC型肝炎の治療効果を予測するためのマーカーを使用して、治療効果を予測する対象となる、具体的な治療方法は、IFN単独療法,PEG化IFN単独療法,PEG化IFN/RBV(2剤併用)療法,PEG化IFN/RBV/その他の薬剤(3剤,あるいは4剤併用)療法等の、IFN又はPEG化IFNを用いた治療等が挙げられる。
本発明のC型肝炎の治療効果を予測するためのマーカーを使用して、治療効果を予測する対象となる、具体的な治療方法は、IFN単独療法,PEG化IFN単独療法,PEG化IFN/RBV(2剤併用)療法,PEG化IFN/RBV/その他の薬剤(3剤,あるいは4剤併用)療法等の、IFN又はPEG化IFNを用いた治療等が挙げられる。
というのも、本発明のマーカーは、2剤併用療法の実際の結果から見いだしたものであるが、これらのマーカーのいくつかが、IL28B蛋白質の遺伝子領域にあり、このIL28Bは、ISG(interferon-stimulated genes)誘導及びTLR(Toll-Like Receptor)を介した抗ウイルス効果を発揮することが報告されていることから(Ank, N. et al. J Immunol 180, 2474-2485 (2008); Marcello, T. et al. Gastroenterology 131, 1887-1898 (2006))、IFNを基軸とする治療全般に影響を与える可能性が高く、IFNを用いた治療方法の効果確認に有用である可能性が非常に高いからである。
本発明のマーカーに含まれる遺伝子多型の中でも、IL28B領域内及びその周辺に存在する、SNP(SNP番号42~55)やその他の遺伝子多型の場合は特に、その可能性が高い。
尚、「PEG化IFN/RBV/その他の薬剤1種又は2種」を用いた3剤併用療法や4剤併用療法は、PEG化IFN/RBV2剤併用療法が基盤となる治療法であり、その意味では、PEG化IFN/RBV2剤併用療法の一種である。
但し、本発明のマーカーが、上述するIL28B関連オリゴ(orポリ)ヌクレオチドその他の、蛋白質をコードする領域を含むものである場合には、IFN以外の、他のC型肝炎治療薬の効果の判定にも使用できる可能性がある。
これらの遺伝子多型のコードする蛋白質自体が、C型肝炎に関係している可能性があるからである。
これらの遺伝子多型のコードする蛋白質自体が、C型肝炎に関係している可能性があるからである。
[本発明のオリゴヌクレオチド若しくはポリヌクレオチド,又はその標識物]
本発明の「オリゴヌクレオチド若しくはポリヌクレオチド,又はその標識物」とは、ヒトゲノム上に存在するオリゴヌクレオチド又はポリヌクレオチドであって上述の遺伝子多型から選択される1種又は2種以上を含むものに、ハイブリダイズするものである。
「上述の遺伝子多型」とは、本発明のマーカーにおいて含まれる各種の遺伝子多型を意味する。
本発明の「オリゴヌクレオチド若しくはポリヌクレオチド,又はその標識物」とは、ヒトゲノム上に存在するオリゴヌクレオチド又はポリヌクレオチドであって上述の遺伝子多型から選択される1種又は2種以上を含むものに、ハイブリダイズするものである。
「上述の遺伝子多型」とは、本発明のマーカーにおいて含まれる各種の遺伝子多型を意味する。
これらのオリゴヌクレオチド若しくはポリヌクレオチド,又はその標識物とは、上記の遺伝子多型を含む領域にハイブリダイズする性質を有することから、C型肝炎の治療効果を予測する際に、患者サンプル中の遺伝子を増幅する際に用いるプライマーや、増幅された遺伝子から、本発明の予測マーカーを釣り上げるためのプローブとして使用できる。
標識物とは、プローブやプライマー等に結合させることによって、目的とするマーカー遺伝子の検出を容易にするものである。
標識物としては、ビオチン,ジゴキシゲニン,放射性標識,酵素標識,蛍光標識等が挙げられる。
標識物としては、ビオチン,ジゴキシゲニン,放射性標識,酵素標識,蛍光標識等が挙げられる。
[本発明のプローブ]
本発明のプローブは、上記本発明のオリゴヌクレオチド若しくはポリヌクレオチド,又はその標識物からなるものである。
本発明のプローブは、上記本発明のオリゴヌクレオチド若しくはポリヌクレオチド,又はその標識物からなるものである。
オリゴヌクレオチド若しくはポリヌクレオチドへの標識物の結合は、公知の方法に従って行うことができる。
《プローブの定義》
プローブとは、特定のオリゴヌクレオチド又はポリヌクレオチド(本件の場合、遺伝子多型を含むマーカーに相当するもの)を釣り上げるのに用いられるものであって、検出の標的となるオリゴ(orポリ)ヌクレオチドと、特異的にハイブリダイズするものを言う。
プローブの配列は、標的の塩基配列と、完全に相補対を形成するものが好ましいが、ハイブリダイズする程度に、完全な相補対と相同性があれば良い。
プローブとは、特定のオリゴヌクレオチド又はポリヌクレオチド(本件の場合、遺伝子多型を含むマーカーに相当するもの)を釣り上げるのに用いられるものであって、検出の標的となるオリゴ(orポリ)ヌクレオチドと、特異的にハイブリダイズするものを言う。
プローブの配列は、標的の塩基配列と、完全に相補対を形成するものが好ましいが、ハイブリダイズする程度に、完全な相補対と相同性があれば良い。
《プローブの役割》
本発明のプローブは、C型肝炎の治療効果を予測する際に、マーカーに相当するオリゴ(orポリ)ヌクレオチドを検出するために用いることができる。
本発明のプローブは、C型肝炎の治療効果を予測する際に、マーカーに相当するオリゴ(orポリ)ヌクレオチドを検出するために用いることができる。
《プローブの決定方法》
プローブは、検出の標的となるオリゴ(orポリ)ヌクレオチド(マーカー)の塩基配列に合わせて、それとハイブリダイズするように、適宜デザインすることができる。
プローブは、検出の標的となるオリゴ(orポリ)ヌクレオチド(マーカー)の塩基配列に合わせて、それとハイブリダイズするように、適宜デザインすることができる。
《プローブの大きさ》
プローブの大きさは、検出の標的となるオリゴ(orポリ)ヌクレオチドによって、様々で一概には言えないが、例えば、~1000塩基長,好ましくは5~500塩基長,より好ましくは10~40塩基長程度とすれば良い。
プローブの大きさは、検出の標的となるオリゴ(orポリ)ヌクレオチドによって、様々で一概には言えないが、例えば、~1000塩基長,好ましくは5~500塩基長,より好ましくは10~40塩基長程度とすれば良い。
尚、通常、プローブとしては、核酸分子の安定性の点等から、DNAが適しているが、リアルタイムPCR法のうち一種のサイクリングプローブ法を用いて標的を増幅する際には、キメラプローブが用いられる。
《プローブの例》
具体的なプローブとしては、例えば、検出の標的であるオリゴ(orポリ)ヌクレオチドが、IL28B関連オリゴ(orポリ)ヌクレオチドである場合には、例えば、下記表3に示したようなものが挙げられる。
具体的なプローブとしては、例えば、検出の標的であるオリゴ(orポリ)ヌクレオチドが、IL28B関連オリゴ(orポリ)ヌクレオチドである場合には、例えば、下記表3に示したようなものが挙げられる。
[本発明のプライマー]
本発明のプライマーは、上記本発明のオリゴヌクレオチド若しくはポリヌクレオチド,又はその標識物からなるものである。
本発明のプライマーは、上記本発明のオリゴヌクレオチド若しくはポリヌクレオチド,又はその標識物からなるものである。
《プライマーの定義》
プライマーとは、特定のオリゴヌクレオチド又はポリヌクレオチド(本件の場合、遺伝子多型を含むマーカーに相当するもの)を増幅する際に用いられるものであって、検出の標的となるオリゴ(orポリ)ヌクレオチドの、アンチセンス鎖及びセンス鎖の3’末端と、それぞれ特異的にハイブリダイズする、センス(フォワード)プライマーとアンチセンス(リバース)プライマーを言う。
プライマーの配列は、標的の塩基配列と、完全に相補対を形成するものが好ましいが、標的とハイブリダイズする程度に、完全な相補対と相同性があれば良い。
プライマーとは、特定のオリゴヌクレオチド又はポリヌクレオチド(本件の場合、遺伝子多型を含むマーカーに相当するもの)を増幅する際に用いられるものであって、検出の標的となるオリゴ(orポリ)ヌクレオチドの、アンチセンス鎖及びセンス鎖の3’末端と、それぞれ特異的にハイブリダイズする、センス(フォワード)プライマーとアンチセンス(リバース)プライマーを言う。
プライマーの配列は、標的の塩基配列と、完全に相補対を形成するものが好ましいが、標的とハイブリダイズする程度に、完全な相補対と相同性があれば良い。
《プライマーの役割》
本発明のプライマーは、C型肝炎の治療効果を予測する際に、サンプル中の、マーカーに相当するオリゴ(orポリ)ヌクレオチドを増幅し、検出を容易にするために用いることができる。
本発明のプライマーは、C型肝炎の治療効果を予測する際に、サンプル中の、マーカーに相当するオリゴ(orポリ)ヌクレオチドを増幅し、検出を容易にするために用いることができる。
《プライマーの決定方法》
プライマーは、検出の標的となるオリゴ(orポリ)ヌクレオチド(マーカー)の末端の塩基配列に合わせて、適宜デザインすることができる。
プライマーは、検出の標的となるオリゴ(orポリ)ヌクレオチド(マーカー)の末端の塩基配列に合わせて、適宜デザインすることができる。
《プライマーの大きさ》
プライマーの大きさは、検出の標的となるオリゴ(orポリ)ヌクレオチドによって、様々で一概には言えないが、例えば、5~100塩基長,好ましくは10~70塩基長,より好ましくは15~50塩基長程度とすれば良い。
プライマーの大きさは、検出の標的となるオリゴ(orポリ)ヌクレオチドによって、様々で一概には言えないが、例えば、5~100塩基長,好ましくは10~70塩基長,より好ましくは15~50塩基長程度とすれば良い。
尚、通常、プライマーとしては、核酸分子の安定性の点等から、DNAが適しているが、等温遺伝子増幅法(ICAN法)にはキメラプライマーが用いられる。
《プライマーの例》
具体的なプライマーとしては、例えば、検出の標的であるオリゴ(orポリ)ヌクレオチドが、IL28B関連オリゴ(orポリ)ヌクレオチドである場合には、例えば、下記表3に示したようなものが挙げられる。
具体的なプライマーとしては、例えば、検出の標的であるオリゴ(orポリ)ヌクレオチドが、IL28B関連オリゴ(orポリ)ヌクレオチドである場合には、例えば、下記表3に示したようなものが挙げられる。
[本発明のC型肝炎の治療効果予測用キット]
本発明のC型肝炎の治療効果予測用キットは、上記本発明のプローブ及び/又はプライマーを含むことを特徴とするものである。
本発明のC型肝炎の治療効果予測用キットは、上記本発明のプローブ及び/又はプライマーを含むことを特徴とするものである。
《キットの役割》
C型肝炎の治療効果予測用キットは、C型肝炎の治療効果を予測するのに用いるキットである。
C型肝炎の治療効果予測用キットは、C型肝炎の治療効果を予測するのに用いるキットである。
《キットの構成要素》
本発明のキットには、プローブ,プライマー,その他のPCRに必要な試薬(耐熱性DNAポリメラーゼ),genotypingに一般に必要とされる、装置・試薬・ソフト・コンピューター等が挙げられ、これらのうち、適宜、必要なものが選択される。
本発明のキットには、プローブ,プライマー,その他のPCRに必要な試薬(耐熱性DNAポリメラーゼ),genotypingに一般に必要とされる、装置・試薬・ソフト・コンピューター等が挙げられ、これらのうち、適宜、必要なものが選択される。
[本発明のC型肝炎の治療効果の予測を行う方法]
本発明のC型肝炎の治療効果の予測を行う方法は、下記(1)及び(2)の工程を有することを特徴とするものである。
本発明のC型肝炎の治療効果の予測を行う方法は、下記(1)及び(2)の工程を有することを特徴とするものである。
(1)患者から得たサンプルにより、下記(X)の遺伝子型を決定する工程。
(X)上述の遺伝子多型の少なくとも1種
(2)(1)で決定された遺伝子型から、C型肝炎の治療効果の予測を行う工程。
(X)上述の遺伝子多型の少なくとも1種
(2)(1)で決定された遺伝子型から、C型肝炎の治療効果の予測を行う工程。
(X)において、「上述の遺伝子多型」とは、本発明のマーカーにおいて含まれる各種の遺伝子多型を意味する。
尚、(X)に含まれる、「配列番号59で表されるポリヌクレオチド内に存在する遺伝子多型」とは、上述したSNP以外の、配列番号59のポリヌクレオチド内に存在する、他の公知の又は未知の、SNPその他の遺伝子多型(多型部分の欠失,置換,付加,及び/又は挿入を含む) を含むものとする。
SNPの集中する配列番号59のポリヌクレオチド領域内の遺伝子多型であれば、C型肝炎の治療効果に関与している可能性が高いからである。
《患者の定義》
患者とは、C型肝炎に感染している,あるいは感染の疑われる患者を意味し、ヒト以外にもチンパンジーやヒト肝細胞置換キメラマウス等を含むものであるが、本発明のマーカーは、ヒトを対象としたGWASによって得られたものであるため、ヒトが最も好ましい。
患者とは、C型肝炎に感染している,あるいは感染の疑われる患者を意味し、ヒト以外にもチンパンジーやヒト肝細胞置換キメラマウス等を含むものであるが、本発明のマーカーは、ヒトを対象としたGWASによって得られたものであるため、ヒトが最も好ましい。
《患者からのサンプル収集方法》
上記の患者から、例えば下記に挙げたようなサンプルを採取する。
上記の患者から、例えば下記に挙げたようなサンプルを採取する。
《サンプルの定義》
サンプルとは、血液,血清,リンパ液,精液,尿,唾液(含:口内粘膜細胞),その他の体液,及び生体組織等を意味するが、血液(リンパ球)が最も採取が容易で、侵襲性が低いという点で好ましい。
サンプルとは、血液,血清,リンパ液,精液,尿,唾液(含:口内粘膜細胞),その他の体液,及び生体組織等を意味するが、血液(リンパ球)が最も採取が容易で、侵襲性が低いという点で好ましい。
《サンプルからのオリゴ(orポリ)ヌクレオチド又は蛋白質の調製》
上記サンプルから、オリゴ(orポリ)ヌクレオチドを調製する。オリゴ(orポリ)ヌクレオチドとしては、ゲノムDNA,又はmRNAを元に逆転写酵素によって調製したcDNA等が挙げられる。
上記サンプルから、オリゴ(orポリ)ヌクレオチドを調製する。オリゴ(orポリ)ヌクレオチドとしては、ゲノムDNA,又はmRNAを元に逆転写酵素によって調製したcDNA等が挙げられる。
尚、遺伝子多型がアミノ酸置換を伴うものである場合には、上記の核酸をもとに得られた蛋白質を用いて遺伝子型の確認を行うことも可能である。
オリゴ(orポリ)ヌクレオチドの調製は、公知の方法に従って行えば良いが、オリゴ(orポリ)ヌクレオチドがDNAの場合には、例えば、サンプルである血液中のリンパ球や顆粒球等の血球細胞から、塩析,PCI(フェノールクロロホルム抽出)法,市販のDNA抽出キット等を用いる方法,その他公知の方法によって行うことができる。
尚、オリゴ(orポリ)ヌクレオチドがmRNAの場合には、PCI法において溶液を酸性にして水層から抽出する方法,オリゴdTカラム等を利用する方法,市販のRNA抽出キットを用いる方法,その他の公知の方法を使用することができる。
《核酸の増幅》
サンプルから得られた核酸の量が少ない場合には、必要に応じて、公知の方法によって増幅することができる。
サンプルから得られた核酸の量が少ない場合には、必要に応じて、公知の方法によって増幅することができる。
核酸がDNAの場合には、PCR法等を用いることができる。
核酸がmRNAの場合には、RT-PCR法を用いることができる。
尚、定量PCR法としては、TaqManプローブやSYBR Green(Power SYBR)を用いたリアルタイムPCR法等も使用することができる。
核酸がmRNAの場合には、RT-PCR法を用いることができる。
尚、定量PCR法としては、TaqManプローブやSYBR Green(Power SYBR)を用いたリアルタイムPCR法等も使用することができる。
《遺伝子型の決定方法》
得られた核酸を用いて、遺伝子多型部分の遺伝子型を決定する。
得られた核酸を用いて、遺伝子多型部分の遺伝子型を決定する。
遺伝子型の決定方法は、公知の配列決定法等を用いることができ、例えば、シークエンサー等を用いて直接塩基配列を決定するダイレクトシークエンス法(ジデオキシ法)の他、RELP(制限酵素断片長多型性)を利用するPCR-RELP法,TaqMan法,DigiTag2法, シングルヌクレオチドプライマー伸長法,SnaPshot法(ABI),ASP-PCR法,PCR-SSO(sequence specific oligonucleotide)法,PCR-SSP(specific sequence primer)法, PCR-SSCP(single-stranded conformational polymorphism analysis)法,電気泳動等による核酸の長さの確認,及びDNAチップ等の遺伝子多型検査用器具等を用いる方法等が挙げられるが、これらに限られるものでは無い。
《遺伝子型による治療効果の予測方法》
上記で得られた遺伝子型の情報を元に、後述する表1~表3等を参照することで、C型肝炎の治療効果を予測することができる。
上記で得られた遺伝子型の情報を元に、後述する表1~表3等を参照することで、C型肝炎の治療効果を予測することができる。
即ち、患者から得たサンプルにおいて、各遺伝子多型のリスクアリルが検出された場合、治療効果が低い可能性が高く、特に、それが、P値の低い遺伝子多型のリスクアリルであればある程、治療効果が低い確率が高いと考えられる。
《本発明の方法によって、治療効果の予測を行う対象となる治療方法》
本発明のC型肝炎の治療効果の予測を行う方法によって治療効果を予測する対象となる、具体的な治療方法は、IFN単独療法,PEG化IFN単独療法,PEG化IFN/RBV(2剤併用)療法,PEG化IFN/RBV/その他の薬剤(3剤,あるいは4剤併用)療法等の、IFN又はPEG化IFNを用いた治療等が挙げられる。
本発明のC型肝炎の治療効果の予測を行う方法によって治療効果を予測する対象となる、具体的な治療方法は、IFN単独療法,PEG化IFN単独療法,PEG化IFN/RBV(2剤併用)療法,PEG化IFN/RBV/その他の薬剤(3剤,あるいは4剤併用)療法等の、IFN又はPEG化IFNを用いた治療等が挙げられる。
但し、本発明のマーカーが、上述するIL28B関連オリゴ(orポリ)ヌクレオチドその他の、蛋白質をコードする領域を含むものである場合には、IFN以外の他のC型肝炎治療薬の効果の判定にも使用できる可能性がある。
これらの遺伝子多型のコードする蛋白質自体が、C型肝炎に関係している可能性があるからである。
これらの遺伝子多型のコードする蛋白質自体が、C型肝炎に関係している可能性があるからである。
[本発明のC型肝炎の治療効果予測マーカーの探索用遺伝子]
本発明の「C型肝炎の治療効果予測マーカーの探索用遺伝子」は、下記の(A)又は(B)の配列を有することを特徴とするものである。
本発明の「C型肝炎の治療効果予測マーカーの探索用遺伝子」は、下記の(A)又は(B)の配列を有することを特徴とするものである。
(A)配列番号59で表されるポリヌクレオチドの少なくとも一部を含むオリゴヌクレオチド又はポリヌクレオチド
(B)(A)のうち、1乃至数個のヌクレオチドが、欠失,置換,付加,及び/又は挿入された、オリゴヌクレオチド又はポリヌクレオチド
(B)(A)のうち、1乃至数個のヌクレオチドが、欠失,置換,付加,及び/又は挿入された、オリゴヌクレオチド又はポリヌクレオチド
配列番号59で表される遺伝子は、L28Bをコードする遺伝子及びその周辺を含む配列(IL28B関連オリゴ(orポリ)ヌクレオチド)である。
これをマーカー探索用遺伝子に選んだのは、上述のSNPのうち多数が、IL28Bを含む当該領域内で見つかったからである。SNPは連鎖することが多いため、C型肝炎の治療効果を左右する未発見の遺伝子多型がこの領域に集中している可能性が非常に高いと考えられるからである。
(B)の変異体をも含めたのは、患者によっては、IL28Bをコードする遺伝子及びその周辺を含む配列が、遺伝子多型やその他の変異等によって、配列番号59のものと多少異なる場合があるからである。
(B)の変異体をも含めたのは、患者によっては、IL28Bをコードする遺伝子及びその周辺を含む配列が、遺伝子多型やその他の変異等によって、配列番号59のものと多少異なる場合があるからである。
「本発明のC型肝炎治療薬のスクリーニング方法」
本発明のC型肝炎治療薬のスクリーニング方法は、下記(A)乃至(C)の少なくとも1種を用いることを特徴とするものである。
本発明のC型肝炎治療薬のスクリーニング方法は、下記(A)乃至(C)の少なくとも1種を用いることを特徴とするものである。
(A)配列番号59で表されるポリヌクレオチドの少なくとも一部を含むオリゴヌクレオチド又はポリヌクレオチド
(B)(A)のうち、1乃至数個のヌクレオチドが、欠失,置換,付加,及び/又は挿入された、オリゴヌクレオチド又はポリヌクレオチド
(C)ヒトゲノム上に存在するオリゴヌクレオチド又はポリヌクレオチドであってrs8103142を含むもの
(B)(A)のうち、1乃至数個のヌクレオチドが、欠失,置換,付加,及び/又は挿入された、オリゴヌクレオチド又はポリヌクレオチド
(C)ヒトゲノム上に存在するオリゴヌクレオチド又はポリヌクレオチドであってrs8103142を含むもの
SNPであるrs8103142の存在している遺伝子のコードする蛋白質(IL28B)自体が、C型肝炎に関係している可能性があることから、IL28B等のrs8103142を含むオリゴ(orポリ)ヌクレオチドが、他のC型肝炎治療薬の効果の判定にも使用できる可能性があるからである。
尚、スクリーニング方法としては、遺伝子や蛋白質の発現量や蛋白質の活性を増減する物質を選択する、各種公知の方法を用いることができ、「in vivo」,「ex vivo」,「cell-free-system」等のいずれの実験系で行っても良い。
「本発明のC型肝炎の予防又は治療剤」
本発明のC型肝炎の予防又は治療剤は、IL28Bタンパク質及び/又はそれをコードする遺伝子を含むことを特徴とするものである。
本発明のC型肝炎の予防又は治療剤は、IL28Bタンパク質及び/又はそれをコードする遺伝子を含むことを特徴とするものである。
本発明のSNPマーカーのマイナーアリル(リスクアリル)をホモ又はヘテロで有する患者(治療効果の低い群)において、メジャーアリルをホモで有する患者(治療効果の高い群)に比べて、有意に、IL28のmRNA発現レベルが低下していることから、IL28Bの発現量も有意に低下している可能性が高く、また上述した通り、IL28B自体が、C型肝炎に関係している可能性が高いこと等から、IL28B又はそれをコードする遺伝子の投与が、C型肝炎の治療に役立つ可能性が高いと考えられるからである。
[GWASによるC型肝炎の治療効果予測]
90万種以上のSNP解析用プローブが搭載されたAffymetrix Genome-Wide Human SNP Array 6.0(以下、SNP Array 6.0)を用いて、C型慢性肝炎に対してPEG-IFN/RBV併用療法が施行された154人のSNPタイピングを行い、薬剤応答性に応じてサブグループ(GWAS Stage:1stパネル:無効(NVR)群82人 vs. 有効(VR)群72人)に分類してゲノムワイド関連解析(GWAS)を行った。
90万種以上のSNP解析用プローブが搭載されたAffymetrix Genome-Wide Human SNP Array 6.0(以下、SNP Array 6.0)を用いて、C型慢性肝炎に対してPEG-IFN/RBV併用療法が施行された154人のSNPタイピングを行い、薬剤応答性に応じてサブグループ(GWAS Stage:1stパネル:無効(NVR)群82人 vs. 有効(VR)群72人)に分類してゲノムワイド関連解析(GWAS)を行った。
P値が10-5レベルあるいはそれ以下のものを選択した結果を表1に示す。
尚、P値は、主にカイ二乗検定によって算出した。
また、無効群とは、治療中ウイルスが一度も消失しなかった例を意味する。
また、無効群とは、治療中ウイルスが一度も消失しなかった例を意味する。
その結果、特に染色体19番目に位置するIL28B 近傍のSNPs (rs12980275 and rs8099917)マイナーアリルが、P値が低く、治療無効に寄与することがわかった(表2)。
(P=1.93×10-13; OR=20.3 and 3.11×10-15; OR=30.0, respectively)。
(P=1.93×10-13; OR=20.3 and 3.11×10-15; OR=30.0, respectively)。
そこで、別のサンプルセットを用いて、下記の14のSNP(配列番号42~55)に関して、検証を行った。
(M)IL28遺伝子近傍のHapMapデータから得られた、上記の2つ(rs12980275 and rs8099917)のSNPsを含む11 SNPs
及び
(N)IL28Bを含む領域のシークエンスにより確認された3 SNPs
及び
(N)IL28Bを含む領域のシークエンスにより確認された3 SNPs
上記のGWAS Stageの154人と、それとは別に、DigiTag2法を用いたReplication Stageとして172人(NVR:50人 vs. VR:122人)の2ndパネルを用い、それらを併せた314人の、combinedのP値を算出し、表3に示した。
合計が314人となっているのは、GWAS Stageの154人中、QCコールレートが95%未満であった12人(NVR:4人+VR:8人)は、GWAS Stageから除かれ、Replication Stageの172人に含められたためである。
合計が314人となっているのは、GWAS Stageの154人中、QCコールレートが95%未満であった12人(NVR:4人+VR:8人)は、GWAS Stageから除かれ、Replication Stageの172人に含められたためである。
QCコールレート(QC call rate)とは、解析可能SNPの割合を意味する。
これらの結果を表3に示す。
表3に示す複数のSNPsが、P = 10-5以下で治療無効に関連していた。
表3に示す複数のSNPsが、P = 10-5以下で治療無効に関連していた。
これらの14SNPsの検証の結果、rs12980275(SNP44),及びrs8099917(SNP50)に加えて、新たに、IL28B に位置する9SNPs (rs955155(SNP42), rs12972991(SNP43), rs8105790(SNP45), rs11881222(SNP46), rs8103142(SNP47), rs28416813(SNP48), rs480321(SNP49), rs7248668(SNP51), rs10853728(SNP52))が、C型肝炎の治療無効に、特に強く関連することが示された。
尚、上記の検証では、GWASデータのValidationおよびReplicationを目的として、DigiTag2法を用いてSNPの遺伝子型を決定した。
DigiTag2法は、解析対象となるSNP毎にSNP特異的なcommon probeと、アリル特異的なquery probeを用いて、一度に複数か所のSNPについて遺伝子型を決定することのできるマルチプレックスSNPタイピング法の一種である(AnalyticalBiochemistry vol.364, No.1, 2007, P.78-85等)。
検出用プローブは、SNP位置と隣接して下流に存在する3'側プローブ(common probe)とSNPを3'末端に含む上流に存在する5'側プローブ'(query probe)に分かれている。
query probeはSNP毎にアリルに対応した2種類を用意する。query probe の3'末端に対応するSNPの遺伝子型に応じて、それと相補的な塩基を持つquery probeだけが、3'側に隣接して存在するcommon probeと結合することができる。
アンチセンス鎖に設計したquery probeは、NCBIデータベースのFasta sequenceと相同な塩基配列となり、センス鎖に設計した場合には相補的な塩基配列となる。
尚、検出のために各query probeの5'末端にはアリルに対応して2種類のquery tagが連結されており、またcommon probeの3'末端にはSNPごとに異なるcommon tagが連結されている。
common probeに連結されたcommon tagと相補的な塩基配列を持つオリゴDNAを固定したDNAチップを用いて検出を行う。検体の遺伝子型に応じて生成されたquery probeとcommon probeの結合産物は、DNAチップ上のcommon tagと相補的な塩基配列を持つオリゴDNAに捕獲される。アリルに対応するquery tagに対応して2種類の蛍光分子を導入することで、各SNPの遺伝子型が決定される。
尚、表2中のaは、Nishida, N. et al. Evaluating the performance of Affymetrix SNP Array 6.0 platform with 400 Japanese individuals. BMC Genomics 9, 431 (2008).から引用した、日本人健常者184人における各SNPのマイナーアリルの頻度(MAF:Minor Allele Frequency)を意味する。
表2中のbは、優性モデルにおける2×2分割表から得たマイナーアリルのOdds ratio(オッズ比)を意味し、ある疾患が起こるリスクを X とするとオッズ比は次のように表される。
Odds=X(1-X)
Odds=X(1-X)
表2中のcは、優性効果モデルにおける、Pearsonのカイ二乗検定によって算出したP値を意味する。
尚、この表3では、アリルは、染色体のセンス鎖で表現している。
そのため、実際にIL28B遺伝子が載っているアンチセンス鎖で表した配列番号59と、この表3とでは、rs11881222, rs8103142, rs28416813, 及びrs4803219の4SNPsのメジャーアリルが、相補的になっている(図2,3の枠で囲った部位参照)。
そのため、実際にIL28B遺伝子が載っているアンチセンス鎖で表した配列番号59と、この表3とでは、rs11881222, rs8103142, rs28416813, 及びrs4803219の4SNPsのメジャーアリルが、相補的になっている(図2,3の枠で囲った部位参照)。
表3中の「SNP type」は、下記を意味する。
gSNP:genomic SNP,発現遺伝子領域以外の領域にあるSNP
iSNP:intron SNP,イントロン領域にあるSNP
nsSNP:nonsynonymous SNP,翻訳領域内にあり、アミノ酸置換を生じるSNP
rSNP:regulatory SNP,発現遺伝子のプロモーター領域にあるSNP
gSNP:genomic SNP,発現遺伝子領域以外の領域にあるSNP
iSNP:intron SNP,イントロン領域にあるSNP
nsSNP:nonsynonymous SNP,翻訳領域内にあり、アミノ酸置換を生じるSNP
rSNP:regulatory SNP,発現遺伝子のプロモーター領域にあるSNP
尚、表3に示す通り、具体的なプライマーやプローブの配列を、配列番号1~58として例示したが、本発明のプライマーやプローブはこれらに限られるものでは無く、これらの配列のうち1乃至数個のヌクレオチドが、欠失,置換,付加,及び/又は挿入されたものであって、本発明のマーカーとハイブリダイズ可能なものも、本発明のプライマーやプローブに含まれることは、言うまでも無い。
また、表3のプライマーやプローブは、配列表にも示した通りDNAであるが、同じ塩基を有するRNAや、DNAとRNAのキメラであっても良い。但し、安定性等の点からDNAが好ましい。
また、表3のプライマーやプローブは、配列表にも示した通りDNAであるが、同じ塩基を有するRNAや、DNAとRNAのキメラであっても良い。但し、安定性等の点からDNAが好ましい。
上述の表1~3等から明らかな通り、本発明のマーカーは、従来のC型肝炎の治療効果予測マーカーに比べても、P値がかなり小さく、予測的中率が高いものであると言うことができる。
その中でも表3で示したSNP番号42~52の11SNPsは特にP値が小さく、中でもSNP45~51の7SNPsは格段にP値が小さく、これらのものは、予測的中率が、極めて高いものである。
その中でも表3で示したSNP番号42~52の11SNPsは特にP値が小さく、中でもSNP45~51の7SNPsは格段にP値が小さく、これらのものは、予測的中率が、極めて高いものである。
[C型肝炎の予防又は治療剤]
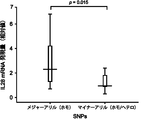
SNP番号42~51のマイナーアリル(リスクアリル)を、ホモあるいはヘテロで有する患者10人(主にPEG-IFN/RBV併用療法において治療が無効であった患者:NVR)及びメジャーアリルをホモで有する患者10人(主にPEG-IFN/RBV併用療法において治療が有効であった患者:VR)の治療前の末梢血単核球から、それぞれRNAを抽出し、リアルタイムRT-PCR法によりIL28 mRNAレベルを検討した結果を、図1に示す。
SNP番号42~51のマイナーアリル(リスクアリル)を、ホモあるいはヘテロで有する患者10人(主にPEG-IFN/RBV併用療法において治療が無効であった患者:NVR)及びメジャーアリルをホモで有する患者10人(主にPEG-IFN/RBV併用療法において治療が有効であった患者:VR)の治療前の末梢血単核球から、それぞれRNAを抽出し、リアルタイムRT-PCR法によりIL28 mRNAレベルを検討した結果を、図1に示す。
図1から分かる通り、マイナーアリルをホモあるいはヘテロで有する群(主にNVR)では、メジャーアリルをホモで有する群(主にVR)に比べて、有意にIL28のmRNA発現レベルが低下していたことから、IL28Bの発現量も有意に低下している可能性が高い。
つまりこのことは、IL28B又はその遺伝子等が、C型肝炎の予防又は治療剤として役立つ可能性が高いことを意味している。
本発明は、より精度の高い、C型肝炎の治療効果の予測を行うことが可能であり、特に「IFN療法」,中でも、昨今の主流となっている「PEG化IFN/RBV併用療法」における、従来にない正確な予測が可能となるため、臨床的な意義は大きい。
(配列番号59においては、小文字は非翻訳領域,大文字は、翻訳領域のDNAを表す。)
配列番号1:sense primer for SNP42: 5' TATAAAGAAAAGGACAGGCCACTTTGGTACGCCAA 3'
配列番号2:antisense primer for SNP42: 5' TAACCTCTCTGAGTACCAGCTTCTTCATTTTCAAGGTAGAA 3'
配列番号3:sense primer for SNP43: 5' TGGGTTGTCCACCTGGACCCAATGTCATCACAAAA 3'
配列番号4:antisense primer for SNP43: 5' GGAGGGAAGGAAGTTCTGACACATGCTACAATAAAAATGAA 3'
配列番号5:sense primer for SNP44,45,46:5' GTCAGCCATCCTCCAATCCCATCAGAGTGGTCTAA 3'
配列番号6:antisense primer for SNP44,45,46:5' TCTCAGGTTGCATGACTGGCGGAAGGGTCAGACACA 3'
配列番号7:sense primer for SNP47,48,49:5' TCAGTTTCTCTTTCCCTCCAGCTGCTCATCTGGCTCA 3'
配列番号8:antisense primer for SNP47,48,49:5' GAGGGACAATGGAGAAGGAGAAGGTGAAGGGGCCA 3'
配列番号9:sense primer for SNP50,51:5' CCAACAAAGTGAGACTGCATCTCTGGGGAAAAAAA 3'
配列番号10:antisense primer for SNP50,51: 5' AGTAACACTTGTTCCTTGTAAAAGATTCCATCCATACAAAAA 3'
配列番号11:sense primer for SNP52:5' CGCAGCTCTGAACAAGATCTTGTTTCTTGGAAAAGTCA 3'
配列番号12:antisense primer for SNP52:5' TGGGTAAAGTAATGAAGCAACAGCCGCTTTCCTAA 3'
配列番号13:sense primer for SNP53:5' TGGCCAACGTGGTGAAACCCCATCTGTACTAAAAATA 3'
配列番号14:antisense primer for SNP53:5' TTCTTACAATTGCACGTGAATCCACAATTATGTCAAAGTA 3'
配列番号15:sense primer for SNP54,55:5' ATATCCTGTTTTACTTACATTGAGTGTGTGTGCAGATGAA 3'
配列番号16:antisense primer for SNP54,55:5' ATACACACACAAAACAGTAGTTGAGAAAAACATGTCTGTA 3'
配列番号17:query probe for SNP42:5' ATTTTGTGTATTTTGTTCTCTGC 3'
配列番号18:query probe for SNP42:5' GAATTTTGTGTATTTTGTTCTCTGT 3'
配列番号19:query probe for SNP43:5' ATGCTGTATGATTCCCCCTA 3'
配列番号20:query probe for SNP43:5' GCTGTATGATTCCCCCTC 3'
配列番号21:query probe for SNP44:5' GAGAAGTCAAATTCCTAGAAACA 3'
配列番号22:query probe for SNP44:5' AGAAGTCAAATTCCTAGAAACG 3'
配列番号23:query probe for SNP45:5' CGGAAGATGTAACCACAGAAG 3'
配列番号24:query probe for SNP45:5' CGGAAGATGTAACCACAGAAA 3'
配列番号25:query probe for SNP46:5' AGCCAGTGTGGTCAGGTA 3'
配列番号26:query probe for SNP46:5' CCAGTGTGGTCAGGTG 3'
配列番号27:query probe for SNP47:5' TCTGCTGAAGGACTGCAG 3'
配列番号28:query probe for SNP47:5' TCTGCTGAAGGACTGCAA 3'
配列番号29:query probe for SNP48:5' GTGGGCAGCCTCTG 3'
配列番号30:query probe for SNP48:5' GTGGGCAGCCTCTC 3'
配列番号31:query probe for SNP49:5' CTGCCCTTCTGTCAGG 3'
配列番号32:query probe for SNP49:5' ACTGCCCTTCTGTCAGA 3'
配列番号33:query probe for SNP50:5' GGTTCCAATTTGGGTGAC 3'
配列番号34:query probe for SNP50:5' GGTTCCAATTTGGGTGAA 3'
配列番号35:query probe for SNP51:5' TGTGCCACTACTATGCTCT 3'
配列番号36:query probe for SNP51:5' TGCCACTACTATGCTCC 3'
配列番号37:query probe for SNP52:5' CATCCTCACCAAAGCTTAG 3'
配列番号38:query probe for SNP52:5' CATCCTCACCAAAGCTTAC 3'
配列番号39:query probe for SNP53:5' CCTAAATATGATTTCCTAAATCATACA 3'
配列番号40:query probe for SNP53:5' CCTAAATATGATTTCCTAAATCATACG 3'
配列番号41:query probe for SNP54:5' TGTCTCAGACGAGCTG 3'
配列番号42:query probe for SNP54:5' GTGTCTCAGACGAGCTA 3'
配列番号43:query probe for SNP55:5' AGAATTATCTGGGCATGC 3'
配列番号44:query probe for SNP55:5' AGAATTATCTGGGCATGG 3'
配列番号45:common probe for SNP42:5' TGGGCCCTCAGTGCACAGGAC 3'
配列番号46:common probe for SNP43:5' CATGAGGTGCTGAGAGAAGTCAAATTCCTAGAAAC 3'
配列番号47:common probe for SNP44:5' GACGTGTCTAAATATTTGCCGGGGTAGCG 3'
配列番号48:common probe for SNP45:5' CAGGACATTGGAGTGATGTCAGGAAGGG 3'
配列番号49:common probe for SNP46:5' GGAGCAGAGGGAAGGGGTAGCAG 3'
配列番号50:common probe for SNP47:5' GTGCCGCTCCCGCCTCTTCC 3'
配列番号51:common probe for SNP48:5' CATTCCCTCAGCTCCCTTTCTCTCTGTGACACA 3'
配列番号52:common probe for SNP49:5' GATAAAAGCTGCCCCATGGAGCTCAGG 3'
配列番号53:common probe for SNP50:5' ATTGCTCACAGAAAGGAAAACAAAAGGAGGAAC 3'
配列番号54:common probe for SNP51:5' AGCTTGGGCTACAGACTGAGACCAT 3'
配列番号55:common probe for SNP52:5' GCCCACATCTCCCAGGCTGCT 3'
配列番号56:common probe for SNP53:5' GACATATTTCCTTGGGAGCTATACATGCATTATGCA 3'
配列番号57:common probe for SNP54:5' TCTCTGGCTTTCATATTGTTATATGAATATAATATTTATT 3'
配列番号58:common probe for SNP55:5' TGGTGGGTGCCTGCAGCTCC 3'
配列番号59:ヒト第19染色体の44425112-44428451位:5'attaagaaatattggcctctgggcatggtggctcacactgaaatcccagcaatttgggaggcctagacagagagatgacttgacatcaggaatttgagaccagccttgccaacatggtgaaacgccatctctactaaaaatataaaaattagctgggaatggtggcacaaatctgtaatctcagctacttgggaggctaaggcaagagaattgcttgaacccaggaggcggaggttgcagttagccaagattttgcactgcactccagcctgggtgaccgaacaagaccctgtctcaaaatatatatatatatatatatatatatatgccaggagtggtggctcaggcctgtaatctcagcactttaataggctgggtgaggaggatggcttgagcccaggagtttgaggctgcagtgagctgtgatcatgccattgcactgcagtgacagagtgagaccctgtcttaaacaacaacaaaaccagagcaggtggaatcctcttgggaacataccttcctgtaggttacccctgagtctccatcagtttctctttccctccagctgctcatctggctcactagccctgccctgctctgggctttcccagcctggggctcccctggtggccggtgtcttacctgaggctgtgttttcacttttcctacatcagctgggactgcccttctgtcagggataaaagctgccccatggagctcaggcaggaattacatcccagacagagctcaaaactgacagaaagagtcaaagccaggacacagtctgagatccagaagaggggactgaaaagaacagagactccagacaagacccaaacagaccctgggtgacagcctcagagtgtttcttctgctgacaaagaccagagatcaggaATGAAACTAGgtgagtcccacatctctgtccgtgctcagctcctgcagcccctgccctcagtgggcagcctctgcattccctcagctccctttctctctgtgacacagACATGACCGGGGACTGCATGCCAGTGCTGGTGCTGATGGCCGCAGTGCTGACCGTGACTGGAGCAGTTCCTGTCGCCAGGCTCCGCGGGGCTCTCCCGGATGCAAGGGGCTGCCACATAGCCCAGTTCAAGTCCCTGTCTCCACAGGAGCTGCAGGCCTTTAAGAGGGCCAAAGATGCCTTAgtgagtctccccctgccctcctgccatggactagcctccacccgcactccaagggtcaccatgctttcccactcccagcttccttcactgggctagcctccaccctccctgcagtgggctatctcatgctcctactgcagggactgactcatgttttcctgaagaagagggtcctctaccatcctcccagcagttaacctcccctatcctgttgtcagccatcctccaatcccatcagagtggtctaacctccacccctcctgctggggctaacctgtgcctttgctgtctagGAAGAGTCGCTTCTGCTGAAGGACTGCAAGTGCCGCTCCCGCCTCTTCCCCAGGACCTGGGACCTGAGGCAGCTGCAGgtgagagggggagtcaggcccacccctgccctcccagccctgctcacctggctctgtagtggccccttcaccttctccttctccattgtccctctctcctctccccacacctgctaccccttccctctgctcctacctgaccacactggctgtgccctctcccctgtgcctgtcaccttcacttgttcctctctatcctcctcccccaacctgttcccctcacctcccccctcacctgctctttctcacctctcctcagGTGAGGGAGCGCCCCGTGGCTTTGGAGGCTGAGCTGGCCCTGACGCTGAAGGTTCTGGAGGCCACCGCTGACACTGACCCAGCCCTGGGGGATGTCTTGGACCAGCCCCTTCACACCCTGCACCATATCCTCTCCCAGCTCCGGGCCTGTgtgagtcgtcagggcccgggcacccaggtctgtgagctctgagcagcgtccttcccctggccaaggccccggctcacacaccgccctcctctgcccacagATCCAGCCTCAGCCCACGGCAGGGCCCAGGACCCGGGGCCGCCTCCACCATTGGCTGCACCGGCTCCAGGAGGCCCCAAAAAAGgtgagtgacccgggaagagagggactgaggtctggggagccactgggagcccagaacccagacagcccctgacccatcccctcctccctacagGAGTCCCCTGGCTGCCTCGAGGCCTCTGTCACCTTCAACCTCTTCCGCCTCCTCACGCGAGACCTGAATTGTGTTGCCAGCGGGGACCTGTGTGTCTGAcccttccgccagtcatgcaacctgagattttatttataaattagccacttggcttaatttattgtcacccagtcgctatttatgtatttgtgtatgtaaatccaactcacctccaggaaaatgtttatttttctactttttgaaatccttgttgaaataaacaatgaggaaaagacacccatgacgtgggactgtgtgtgcgttggtgtgtatttcctttgcattgctgccataacaaattaccctaaaagtagcatctagaacagcaggttcattgagtctgtgctgtccactggggtccccaggtcacatgtcactatcgagcacctggaatgtaggtggtgcaacctcattttttcagtgagtgtaaaacataccccagaatttgaagatttactgtggagaaaaaaaaaaaagtaaaacagcttaattataatttatatattcattagatgttgaaatgatagcactttggatgtattgggtgaaatgaagcatctttaaaattaatttagagcctggatgtgattgctcaagcctgtaatcacagcactttgggtggctgagttgggaggatcacttgagcccagaccagcctgggcaacatagcaagatcctgtctctataaaaaattagagaattggctgggtgtggtggcacaagcccataatcccagctactggtggaagctgagatgggaggactgtttgaggtcaggagttcacgaccagcctgagcaacagcaagatcctgtctcttgcgtgatcacatctcactgtagcctcaaactcctgggctcaagtgattctctctcctcggcaccatcctccaccctctccagcccctccccgaactgggattacaggcttgagtcaccgggtccagcccattctctttccaagaagcccctgaattgtataagcaaacatgaggtctcactaaccctggataagcccctacagatgaggaggacaacttggatcagggacccctgctgagt 3'
配列番号1:sense primer for SNP42: 5' TATAAAGAAAAGGACAGGCCACTTTGGTACGCCAA 3'
配列番号2:antisense primer for SNP42: 5' TAACCTCTCTGAGTACCAGCTTCTTCATTTTCAAGGTAGAA 3'
配列番号3:sense primer for SNP43: 5' TGGGTTGTCCACCTGGACCCAATGTCATCACAAAA 3'
配列番号4:antisense primer for SNP43: 5' GGAGGGAAGGAAGTTCTGACACATGCTACAATAAAAATGAA 3'
配列番号5:sense primer for SNP44,45,46:5' GTCAGCCATCCTCCAATCCCATCAGAGTGGTCTAA 3'
配列番号6:antisense primer for SNP44,45,46:5' TCTCAGGTTGCATGACTGGCGGAAGGGTCAGACACA 3'
配列番号7:sense primer for SNP47,48,49:5' TCAGTTTCTCTTTCCCTCCAGCTGCTCATCTGGCTCA 3'
配列番号8:antisense primer for SNP47,48,49:5' GAGGGACAATGGAGAAGGAGAAGGTGAAGGGGCCA 3'
配列番号9:sense primer for SNP50,51:5' CCAACAAAGTGAGACTGCATCTCTGGGGAAAAAAA 3'
配列番号10:antisense primer for SNP50,51: 5' AGTAACACTTGTTCCTTGTAAAAGATTCCATCCATACAAAAA 3'
配列番号11:sense primer for SNP52:5' CGCAGCTCTGAACAAGATCTTGTTTCTTGGAAAAGTCA 3'
配列番号12:antisense primer for SNP52:5' TGGGTAAAGTAATGAAGCAACAGCCGCTTTCCTAA 3'
配列番号13:sense primer for SNP53:5' TGGCCAACGTGGTGAAACCCCATCTGTACTAAAAATA 3'
配列番号14:antisense primer for SNP53:5' TTCTTACAATTGCACGTGAATCCACAATTATGTCAAAGTA 3'
配列番号15:sense primer for SNP54,55:5' ATATCCTGTTTTACTTACATTGAGTGTGTGTGCAGATGAA 3'
配列番号16:antisense primer for SNP54,55:5' ATACACACACAAAACAGTAGTTGAGAAAAACATGTCTGTA 3'
配列番号17:query probe for SNP42:5' ATTTTGTGTATTTTGTTCTCTGC 3'
配列番号18:query probe for SNP42:5' GAATTTTGTGTATTTTGTTCTCTGT 3'
配列番号19:query probe for SNP43:5' ATGCTGTATGATTCCCCCTA 3'
配列番号20:query probe for SNP43:5' GCTGTATGATTCCCCCTC 3'
配列番号21:query probe for SNP44:5' GAGAAGTCAAATTCCTAGAAACA 3'
配列番号22:query probe for SNP44:5' AGAAGTCAAATTCCTAGAAACG 3'
配列番号23:query probe for SNP45:5' CGGAAGATGTAACCACAGAAG 3'
配列番号24:query probe for SNP45:5' CGGAAGATGTAACCACAGAAA 3'
配列番号25:query probe for SNP46:5' AGCCAGTGTGGTCAGGTA 3'
配列番号26:query probe for SNP46:5' CCAGTGTGGTCAGGTG 3'
配列番号27:query probe for SNP47:5' TCTGCTGAAGGACTGCAG 3'
配列番号28:query probe for SNP47:5' TCTGCTGAAGGACTGCAA 3'
配列番号29:query probe for SNP48:5' GTGGGCAGCCTCTG 3'
配列番号30:query probe for SNP48:5' GTGGGCAGCCTCTC 3'
配列番号31:query probe for SNP49:5' CTGCCCTTCTGTCAGG 3'
配列番号32:query probe for SNP49:5' ACTGCCCTTCTGTCAGA 3'
配列番号33:query probe for SNP50:5' GGTTCCAATTTGGGTGAC 3'
配列番号34:query probe for SNP50:5' GGTTCCAATTTGGGTGAA 3'
配列番号35:query probe for SNP51:5' TGTGCCACTACTATGCTCT 3'
配列番号36:query probe for SNP51:5' TGCCACTACTATGCTCC 3'
配列番号37:query probe for SNP52:5' CATCCTCACCAAAGCTTAG 3'
配列番号38:query probe for SNP52:5' CATCCTCACCAAAGCTTAC 3'
配列番号39:query probe for SNP53:5' CCTAAATATGATTTCCTAAATCATACA 3'
配列番号40:query probe for SNP53:5' CCTAAATATGATTTCCTAAATCATACG 3'
配列番号41:query probe for SNP54:5' TGTCTCAGACGAGCTG 3'
配列番号42:query probe for SNP54:5' GTGTCTCAGACGAGCTA 3'
配列番号43:query probe for SNP55:5' AGAATTATCTGGGCATGC 3'
配列番号44:query probe for SNP55:5' AGAATTATCTGGGCATGG 3'
配列番号45:common probe for SNP42:5' TGGGCCCTCAGTGCACAGGAC 3'
配列番号46:common probe for SNP43:5' CATGAGGTGCTGAGAGAAGTCAAATTCCTAGAAAC 3'
配列番号47:common probe for SNP44:5' GACGTGTCTAAATATTTGCCGGGGTAGCG 3'
配列番号48:common probe for SNP45:5' CAGGACATTGGAGTGATGTCAGGAAGGG 3'
配列番号49:common probe for SNP46:5' GGAGCAGAGGGAAGGGGTAGCAG 3'
配列番号50:common probe for SNP47:5' GTGCCGCTCCCGCCTCTTCC 3'
配列番号51:common probe for SNP48:5' CATTCCCTCAGCTCCCTTTCTCTCTGTGACACA 3'
配列番号52:common probe for SNP49:5' GATAAAAGCTGCCCCATGGAGCTCAGG 3'
配列番号53:common probe for SNP50:5' ATTGCTCACAGAAAGGAAAACAAAAGGAGGAAC 3'
配列番号54:common probe for SNP51:5' AGCTTGGGCTACAGACTGAGACCAT 3'
配列番号55:common probe for SNP52:5' GCCCACATCTCCCAGGCTGCT 3'
配列番号56:common probe for SNP53:5' GACATATTTCCTTGGGAGCTATACATGCATTATGCA 3'
配列番号57:common probe for SNP54:5' TCTCTGGCTTTCATATTGTTATATGAATATAATATTTATT 3'
配列番号58:common probe for SNP55:5' TGGTGGGTGCCTGCAGCTCC 3'
配列番号59:ヒト第19染色体の44425112-44428451位:5'attaagaaatattggcctctgggcatggtggctcacactgaaatcccagcaatttgggaggcctagacagagagatgacttgacatcaggaatttgagaccagccttgccaacatggtgaaacgccatctctactaaaaatataaaaattagctgggaatggtggcacaaatctgtaatctcagctacttgggaggctaaggcaagagaattgcttgaacccaggaggcggaggttgcagttagccaagattttgcactgcactccagcctgggtgaccgaacaagaccctgtctcaaaatatatatatatatatatatatatatatgccaggagtggtggctcaggcctgtaatctcagcactttaataggctgggtgaggaggatggcttgagcccaggagtttgaggctgcagtgagctgtgatcatgccattgcactgcagtgacagagtgagaccctgtcttaaacaacaacaaaaccagagcaggtggaatcctcttgggaacataccttcctgtaggttacccctgagtctccatcagtttctctttccctccagctgctcatctggctcactagccctgccctgctctgggctttcccagcctggggctcccctggtggccggtgtcttacctgaggctgtgttttcacttttcctacatcagctgggactgcccttctgtcagggataaaagctgccccatggagctcaggcaggaattacatcccagacagagctcaaaactgacagaaagagtcaaagccaggacacagtctgagatccagaagaggggactgaaaagaacagagactccagacaagacccaaacagaccctgggtgacagcctcagagtgtttcttctgctgacaaagaccagagatcaggaATGAAACTAGgtgagtcccacatctctgtccgtgctcagctcctgcagcccctgccctcagtgggcagcctctgcattccctcagctccctttctctctgtgacacagACATGACCGGGGACTGCATGCCAGTGCTGGTGCTGATGGCCGCAGTGCTGACCGTGACTGGAGCAGTTCCTGTCGCCAGGCTCCGCGGGGCTCTCCCGGATGCAAGGGGCTGCCACATAGCCCAGTTCAAGTCCCTGTCTCCACAGGAGCTGCAGGCCTTTAAGAGGGCCAAAGATGCCTTAgtgagtctccccctgccctcctgccatggactagcctccacccgcactccaagggtcaccatgctttcccactcccagcttccttcactgggctagcctccaccctccctgcagtgggctatctcatgctcctactgcagggactgactcatgttttcctgaagaagagggtcctctaccatcctcccagcagttaacctcccctatcctgttgtcagccatcctccaatcccatcagagtggtctaacctccacccctcctgctggggctaacctgtgcctttgctgtctagGAAGAGTCGCTTCTGCTGAAGGACTGCAAGTGCCGCTCCCGCCTCTTCCCCAGGACCTGGGACCTGAGGCAGCTGCAGgtgagagggggagtcaggcccacccctgccctcccagccctgctcacctggctctgtagtggccccttcaccttctccttctccattgtccctctctcctctccccacacctgctaccccttccctctgctcctacctgaccacactggctgtgccctctcccctgtgcctgtcaccttcacttgttcctctctatcctcctcccccaacctgttcccctcacctcccccctcacctgctctttctcacctctcctcagGTGAGGGAGCGCCCCGTGGCTTTGGAGGCTGAGCTGGCCCTGACGCTGAAGGTTCTGGAGGCCACCGCTGACACTGACCCAGCCCTGGGGGATGTCTTGGACCAGCCCCTTCACACCCTGCACCATATCCTCTCCCAGCTCCGGGCCTGTgtgagtcgtcagggcccgggcacccaggtctgtgagctctgagcagcgtccttcccctggccaaggccccggctcacacaccgccctcctctgcccacagATCCAGCCTCAGCCCACGGCAGGGCCCAGGACCCGGGGCCGCCTCCACCATTGGCTGCACCGGCTCCAGGAGGCCCCAAAAAAGgtgagtgacccgggaagagagggactgaggtctggggagccactgggagcccagaacccagacagcccctgacccatcccctcctccctacagGAGTCCCCTGGCTGCCTCGAGGCCTCTGTCACCTTCAACCTCTTCCGCCTCCTCACGCGAGACCTGAATTGTGTTGCCAGCGGGGACCTGTGTGTCTGAcccttccgccagtcatgcaacctgagattttatttataaattagccacttggcttaatttattgtcacccagtcgctatttatgtatttgtgtatgtaaatccaactcacctccaggaaaatgtttatttttctactttttgaaatccttgttgaaataaacaatgaggaaaagacacccatgacgtgggactgtgtgtgcgttggtgtgtatttcctttgcattgctgccataacaaattaccctaaaagtagcatctagaacagcaggttcattgagtctgtgctgtccactggggtccccaggtcacatgtcactatcgagcacctggaatgtaggtggtgcaacctcattttttcagtgagtgtaaaacataccccagaatttgaagatttactgtggagaaaaaaaaaaaagtaaaacagcttaattataatttatatattcattagatgttgaaatgatagcactttggatgtattgggtgaaatgaagcatctttaaaattaatttagagcctggatgtgattgctcaagcctgtaatcacagcactttgggtggctgagttgggaggatcacttgagcccagaccagcctgggcaacatagcaagatcctgtctctataaaaaattagagaattggctgggtgtggtggcacaagcccataatcccagctactggtggaagctgagatgggaggactgtttgaggtcaggagttcacgaccagcctgagcaacagcaagatcctgtctcttgcgtgatcacatctcactgtagcctcaaactcctgggctcaagtgattctctctcctcggcaccatcctccaccctctccagcccctccccgaactgggattacaggcttgagtcaccgggtccagcccattctctttccaagaagcccctgaattgtataagcaaacatgaggtctcactaaccctggataagcccctacagatgaggaggacaacttggatcagggacccctgctgagt 3'
Claims (14)
- ヒトゲノム上に存在するオリゴヌクレオチド又はポリヌクレオチドであって、下記に示す遺伝子多型,又はこれらと連鎖不平衡状態(R2>0.8)にあるものから選択される1種又は2種以上を含むものであることを特徴とする、C型肝炎の治療効果を予測するためのマーカー。
rs12043519, rs1986953, rs13392065, rs16987149, rs16987155, rs2374341, rs2374342, rs7566701, rs12713624, rs10208788, rs10208883, rs9876122, rs2036081, rs13122167, rs4488950, rs13150115, rs7663113, rs1379606, rs913500, rs6458003, rs6906840, rs2224068, rs9390164, rs4512312, rs757844, rs4876271, rs7942675, rs17787293, rs917334, rs6489019, rs6489020, rs6489021, rs11610391, rs9528038, rs341554, rs2325219, rs1353348, rs2076954, rs12907066, rs8075713, rs4795794, rs955155, rs12972991, rs12980275, rs8105790, rs11881222, rs8103142, rs28416813, rs4803219, rs8099917, rs7248668, rs10853728, rs4803223, rs12980602,rs4803224,配列番号59で表されるポリヌクレオチド内に存在する遺伝子多型 - オリゴヌクレオチド又はポリヌクレオチドの長さが、40k塩基長以下であることを特徴とする、請求項1記載の、C型肝炎の治療効果を予測するためのマーカー。
- C型肝炎の治療効果が、インターフェロン及び/又はPEG化インターフェロンを用いた治療による治療効果であることを特徴とする、請求項1又は2に記載の、C型肝炎の治療効果を予測するためのマーカー。
- C型肝炎の治療効果が、PEG化インターフェロン/RBV併用療法による治療効果であることを特徴とする、請求項1乃至3のいずれかに記載の、C型肝炎の治療効果を予測するためのマーカー。
- ヒトゲノム上に存在するオリゴヌクレオチド又はポリヌクレオチドであって請求項1記載の遺伝子多型から選択される1種又は2種以上を含むものに、ハイブリダイズするオリゴヌクレオチド若しくはポリヌクレオチド,又はその標識物。
- 請求項5記載のオリゴヌクレオチド若しくはポリヌクレオチド,又はその標識物からなるプローブ。
- 請求項5記載のオリゴヌクレオチド若しくはポリヌクレオチド,又はその標識物からなるプライマー。
- 請求項6記載のプローブ及び/又は請求項7記載のプライマーを含むことを特徴とする、C型肝炎の治療効果予測用キット。
- 下記(1)及び(2)の工程を有することを特徴とする、C型肝炎の治療効果の予測を行う方法。
(1)患者から得たサンプルにより、下記(X)の遺伝子型を決定する工程。
(X)請求項1記載の遺伝子多型の少なくとも1種
(2)(1)で決定された遺伝子型から、C型肝炎の治療効果の予測を行う工程。 - C型肝炎の治療効果が、インターフェロン及び/又はPEG化インターフェロンを用いた治療による治療効果であることを特徴とする、請求項9記載の、C型肝炎の治療効果の予測を行う方法。
- C型肝炎の治療効果が、PEG化インターフェロン/RBV併用療法による治療効果であることを特徴とする、請求項10記載の、C型肝炎の治療効果の予測を行う方法。
- 下記の(A)又は(B)の配列を有することを特徴とする、C型肝炎の治療効果予測マーカーの探索用遺伝子。
(A)配列番号59で表されるポリヌクレオチドの少なくとも一部を含むオリゴヌクレオチド又はポリヌクレオチド
(B)(A)のうち、1乃至数個のヌクレオチドが、欠失,置換,付加,及び/又は挿入された、オリゴヌクレオチド又はポリヌクレオチド - 下記(A)乃至(C)の少なくとも1種を用いることを特徴とする、C型肝炎治療薬のスクリーニング方法。
(A)配列番号59で表されるポリヌクレオチドの少なくとも一部を含むオリゴヌクレオチド又はポリヌクレオチド
(B)(A)のうち、1乃至数個のヌクレオチドが、欠失,置換,付加,及び/又は挿入された、オリゴヌクレオチド又はポリヌクレオチド
(C)ヒトゲノム上に存在するオリゴヌクレオチド又はポリヌクレオチドであってrs8103142を含むもの - IL28Bタンパク質及び/又はそれをコードする遺伝子を含むことを特徴とする、C型肝炎の予防又は治療剤。
Priority Applications (1)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| CN2010800365956A CN102482664A (zh) | 2009-08-21 | 2010-08-05 | 用于预测c型肝炎的治疗效果的标记物和预测c型肝炎的治疗效果的方法以及c型肝炎的预防或治疗剂 |
Applications Claiming Priority (2)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| JP2009-192615 | 2009-08-21 | ||
| JP2009192615A JP5727694B2 (ja) | 2009-08-21 | 2009-08-21 | C型肝炎の治療効果を予測するためのマーカー及びc型肝炎の治療効果の予測を行う方法並びにc型肝炎の予防又は治療剤 |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| WO2011021508A1 true WO2011021508A1 (ja) | 2011-02-24 |
Family
ID=43606963
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| PCT/JP2010/063262 WO2011021508A1 (ja) | 2009-08-21 | 2010-08-05 | C型肝炎の治療効果を予測するためのマーカー及びc型肝炎の治療効果の予測を行う方法並びにc型肝炎の予防又は治療剤 |
Country Status (3)
| Country | Link |
|---|---|
| JP (1) | JP5727694B2 (ja) |
| CN (1) | CN102482664A (ja) |
| WO (1) | WO2011021508A1 (ja) |
Cited By (3)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2011115033A1 (ja) * | 2010-03-19 | 2011-09-22 | 財団法人ヒューマンサイエンス振興財団 | C型肝炎の治療効果を予測するためのマーカー群、検査方法及び検査用キット |
| CN103074417A (zh) * | 2012-06-20 | 2013-05-01 | 海尔施生物医药股份有限公司 | 一种丙型肝炎治疗用药指导的多重基因检测方法 |
| CN105749246A (zh) * | 2011-03-31 | 2016-07-13 | 诺华股份有限公司 | 治疗丙肝病毒感染的阿拉泊韦 |
Families Citing this family (4)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| JP5656159B2 (ja) * | 2009-08-24 | 2015-01-21 | 独立行政法人理化学研究所 | インターフェロン療法の効果予測用マーカー |
| CN102816838A (zh) * | 2012-07-06 | 2012-12-12 | 吉林艾迪康医学检验所有限公司 | 用于检测丙型肝炎患者il28b snp12980275多态性的试剂盒 |
| EP3012326A4 (en) | 2013-03-26 | 2016-12-21 | Nippon Gene Co Ltd | PRIMER AND PROBE SET FOR IDENTIFYING GENPOLYMORPHISM AND USE THEREOF |
| JP6155750B2 (ja) * | 2013-03-27 | 2017-07-05 | 東洋紡株式会社 | SNP(rs11881222)を検出するためのプローブ |
-
2009
- 2009-08-21 JP JP2009192615A patent/JP5727694B2/ja active Active
-
2010
- 2010-08-05 CN CN2010800365956A patent/CN102482664A/zh active Pending
- 2010-08-05 WO PCT/JP2010/063262 patent/WO2011021508A1/ja active Application Filing
Non-Patent Citations (5)
| Title |
|---|
| GE D. ET AL: "Genetic variation in IL28B predicts hepatitis C treatment-induced viral clearance", NATURE, vol. 461, no. 7262, 16 August 2009 (2009-08-16), pages 399 - 401, XP002602519, DOI: doi:10.1038/NATURE08309 * |
| MORGAN T. R. ET AL: "DNA polymorphisms and response to treatment in patients with chronic hepatitis C: results from the HALT-C trial", J. HEPATOL., vol. 49, no. 4, 5 May 2008 (2008-05-05), pages 548 - 556, XP025432161, DOI: doi:10.1016/j.jhep.2008.05.011 * |
| SELZNER N. ET AL: "Can genetic variations predict HCV treatment outcomes?", J. HEPATOL., vol. 49, no. 4, 21 July 2008 (2008-07-21), pages 494 - 497, XP025432153, DOI: doi:10.1016/j.jhep.2008.07.006 * |
| SUPPIAH V. ET AL: "IL28B is associated with response to chronic hepatitis C interferon-alpha and ribavirin therapy", NAT. GENET., vol. 41, no. 10, 13 September 2009 (2009-09-13), pages 1100 - 1104, XP002601552, DOI: doi:10.1038/NG.447 * |
| TANAKA Y. ET AL: "Genome-wide association of IL28B with response to pegylated interferon-alpha and ribavirin therapy for chronic hepatitis C", NAT. GENET., vol. 41, no. 10, 13 September 2009 (2009-09-13), pages 1105 - 1109, XP002601553, DOI: doi:10.1038/NG.449 * |
Cited By (4)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2011115033A1 (ja) * | 2010-03-19 | 2011-09-22 | 財団法人ヒューマンサイエンス振興財団 | C型肝炎の治療効果を予測するためのマーカー群、検査方法及び検査用キット |
| JP2011193786A (ja) * | 2010-03-19 | 2011-10-06 | Japan Health Science Foundation | C型肝炎の治療効果を予測するためのマーカー群、検査方法及び検査用キット |
| CN105749246A (zh) * | 2011-03-31 | 2016-07-13 | 诺华股份有限公司 | 治疗丙肝病毒感染的阿拉泊韦 |
| CN103074417A (zh) * | 2012-06-20 | 2013-05-01 | 海尔施生物医药股份有限公司 | 一种丙型肝炎治疗用药指导的多重基因检测方法 |
Also Published As
| Publication number | Publication date |
|---|---|
| JP2011041526A (ja) | 2011-03-03 |
| CN102482664A (zh) | 2012-05-30 |
| JP5727694B2 (ja) | 2015-06-03 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP5727694B2 (ja) | C型肝炎の治療効果を予測するためのマーカー及びc型肝炎の治療効果の予測を行う方法並びにc型肝炎の予防又は治療剤 | |
| KR101633783B1 (ko) | Cyp3a5 및 cyp3a7 유전자 부위의 염기서열 다형성을 이용한 타크로리무스 혈중 약물 농도 예측 방법 | |
| EP1888777A2 (en) | Methods and compositions for assessment of pulmonary function and disorders | |
| EP2937416B1 (en) | Identification of group of hypertension-susceptibility genes | |
| EP2821503B1 (en) | Method for detecting hla-a*31:01 allele | |
| EP3008206B1 (en) | Method for detecting the susceptibility of a dog to hip dysplasia | |
| US20160076104A1 (en) | Methods and compositions for assessment of pulmonary function and disorders | |
| JP2013070660A (ja) | C型肝炎患者の経過予測方法 | |
| CA2673092A1 (en) | Methods and compositions for the assessment of cardiovascular function and disorders | |
| US20050266410A1 (en) | Methods of Human Leukocyte Antigen typing by neighboring single nucleotide polymorphism haplotypes | |
| WO2001053537A2 (en) | Nitric oxide synthase gene diagnostic polymorphisms | |
| CN115679003A (zh) | 用于预测药物疗效的组合物、系统及用途 | |
| KR101100437B1 (ko) | 단일염기 다형을 포함하는 대장암과 관련된 폴리뉴클레오티드, 그를 포함하는 마이크로어레이 및 진단 키트 및 그를 이용한 대장암의 진단방법 | |
| JP5787337B2 (ja) | C型肝炎の治療効果を予測するためのマーカー群、検査方法及び検査用キット | |
| CN110029162B (zh) | 一种用于检测系统性红斑狼疮易感性位于非编码基因区的snp标志物及其应用 | |
| US20130281319A1 (en) | Methods and compositions for assessment of pulmonary function and disorders | |
| KR101174823B1 (ko) | 개인식별용 단일염기다형성 마커 및 그의 용도 | |
| JP6245796B2 (ja) | 原発性胆汁性肝硬変の発症リスク予測マーカー、プローブ、プライマー及びキット並びに原発性胆汁性肝硬変の発症リスク予測方法 | |
| CN105765077B (zh) | 测定抗甲状腺药物诱导的粒细胞缺乏症风险的检测方法以及测定用试剂盒 | |
| JP5416962B2 (ja) | 薬剤による光線過敏症の発症の危険性の判定方法 | |
| EP2566978A1 (en) | Antiviral treatment susceptibility gene and uses thereof | |
| JP5070552B2 (ja) | C型慢性肝炎治療効果予測のための検査方法及び検査用キット | |
| CN115976184A (zh) | 用于预测干扰素α对乙型肝炎患者治疗疗效的分子标记及其应用 | |
| KR100695147B1 (ko) | 다중좌 마커를 이용한 2형 당뇨병의 진단방법, 2형당뇨병과 연관된 마커를 포함하는 폴리뉴클레오티드 및마이크로어레이 | |
| KR101168737B1 (ko) | Fanca 유전자로부터 유래된 단일염기다형을 포함하는 폴리뉴클레오티드, 이를 포함하는 마이크로어레이 및 진단키트, 및 이를 이용한 분석방법 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| WWE | Wipo information: entry into national phase |
Ref document number: 201080036595.6 Country of ref document: CN |
|
| 121 | Ep: the epo has been informed by wipo that ep was designated in this application |
Ref document number: 10809853 Country of ref document: EP Kind code of ref document: A1 |
|
| NENP | Non-entry into the national phase |
Ref country code: DE |
|
| 122 | Ep: pct application non-entry in european phase |
Ref document number: 10809853 Country of ref document: EP Kind code of ref document: A1 |