ES2874875T3 - Procedimiento de tratamiento de adenocarcinoma de pulmón - Google Patents
Procedimiento de tratamiento de adenocarcinoma de pulmón Download PDFInfo
- Publication number
- ES2874875T3 ES2874875T3 ES15721100T ES15721100T ES2874875T3 ES 2874875 T3 ES2874875 T3 ES 2874875T3 ES 15721100 T ES15721100 T ES 15721100T ES 15721100 T ES15721100 T ES 15721100T ES 2874875 T3 ES2874875 T3 ES 2874875T3
- Authority
- ES
- Spain
- Prior art keywords
- ros1
- compound
- fusion
- slc34a2
- present disclosure
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
- 238000000034 method Methods 0.000 title claims abstract description 152
- 201000005249 lung adenocarcinoma Diseases 0.000 title claims abstract description 47
- 208000010507 Adenocarcinoma of Lung Diseases 0.000 title claims abstract description 45
- 238000011282 treatment Methods 0.000 title claims description 112
- 230000004927 fusion Effects 0.000 claims abstract description 326
- 150000001875 compounds Chemical class 0.000 claims abstract description 195
- 150000003839 salts Chemical class 0.000 claims abstract description 117
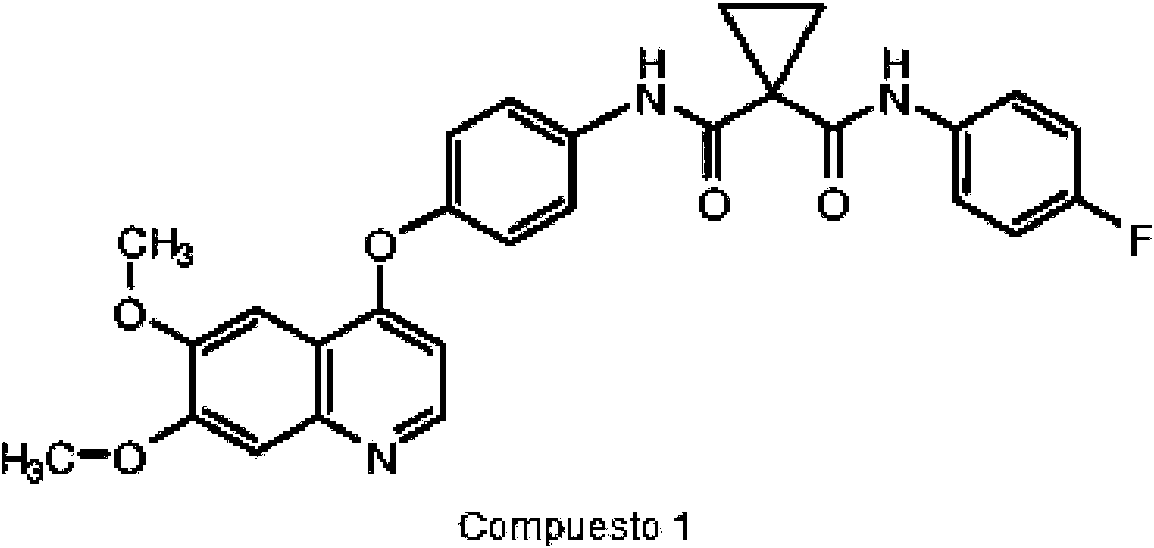
- 229940125904 compound 1 Drugs 0.000 claims abstract description 105
- 208000002154 non-small cell lung carcinoma Diseases 0.000 claims abstract description 95
- 208000029729 tumor suppressor gene on chromosome 11 Diseases 0.000 claims abstract description 92
- 101000686031 Homo sapiens Proto-oncogene tyrosine-protein kinase ROS Proteins 0.000 claims description 349
- 102100023347 Proto-oncogene tyrosine-protein kinase ROS Human genes 0.000 claims description 329
- 206010028980 Neoplasm Diseases 0.000 claims description 105
- 108090000765 processed proteins & peptides Proteins 0.000 claims description 95
- 229920001184 polypeptide Polymers 0.000 claims description 92
- 102000004196 processed proteins & peptides Human genes 0.000 claims description 92
- 201000011510 cancer Diseases 0.000 claims description 87
- 239000012634 fragment Substances 0.000 claims description 71
- KTEIFNKAUNYNJU-GFCCVEGCSA-N crizotinib Chemical compound O([C@H](C)C=1C(=C(F)C=CC=1Cl)Cl)C(C(=NC=1)N)=CC=1C(=C1)C=NN1C1CCNCC1 KTEIFNKAUNYNJU-GFCCVEGCSA-N 0.000 claims description 47
- 239000002146 L01XE16 - Crizotinib Substances 0.000 claims description 43
- 229960005061 crizotinib Drugs 0.000 claims description 41
- 238000001514 detection method Methods 0.000 claims description 39
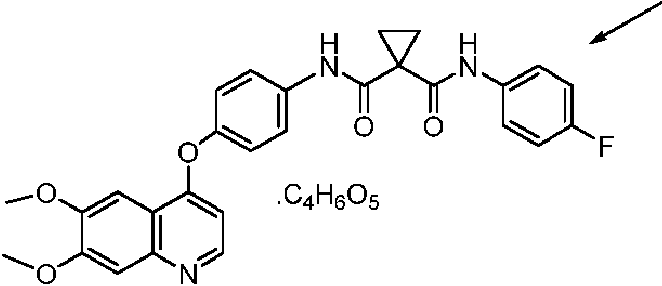
- 150000004701 malic acid derivatives Chemical class 0.000 claims description 38
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 claims description 32
- 239000002775 capsule Substances 0.000 claims description 28
- 239000012458 free base Substances 0.000 claims description 27
- SDUQYLNIPVEERB-QPPQHZFASA-N gemcitabine Chemical compound O=C1N=C(N)C=CN1[C@H]1C(F)(F)[C@H](O)[C@@H](CO)O1 SDUQYLNIPVEERB-QPPQHZFASA-N 0.000 claims description 27
- 229960005277 gemcitabine Drugs 0.000 claims description 27
- AICOOMRHRUFYCM-ZRRPKQBOSA-N oxazine, 1 Chemical compound C([C@@H]1[C@H](C(C[C@]2(C)[C@@H]([C@H](C)N(C)C)[C@H](O)C[C@]21C)=O)CC1=CC2)C[C@H]1[C@@]1(C)[C@H]2N=C(C(C)C)OC1 AICOOMRHRUFYCM-ZRRPKQBOSA-N 0.000 claims description 25
- VSRXQHXAPYXROS-UHFFFAOYSA-N azanide;cyclobutane-1,1-dicarboxylic acid;platinum(2+) Chemical compound [NH2-].[NH2-].[Pt+2].OC(=O)C1(C(O)=O)CCC1 VSRXQHXAPYXROS-UHFFFAOYSA-N 0.000 claims description 23
- 229960004562 carboplatin Drugs 0.000 claims description 23
- 201000010099 disease Diseases 0.000 claims description 22
- ZDZOTLJHXYCWBA-VCVYQWHSSA-N N-debenzoyl-N-(tert-butoxycarbonyl)-10-deacetyltaxol Chemical compound O([C@H]1[C@H]2[C@@](C([C@H](O)C3=C(C)[C@@H](OC(=O)[C@H](O)[C@@H](NC(=O)OC(C)(C)C)C=4C=CC=CC=4)C[C@]1(O)C3(C)C)=O)(C)[C@@H](O)C[C@H]1OC[C@]12OC(=O)C)C(=O)C1=CC=CC=C1 ZDZOTLJHXYCWBA-VCVYQWHSSA-N 0.000 claims description 21
- 229960003668 docetaxel Drugs 0.000 claims description 21
- 239000008194 pharmaceutical composition Substances 0.000 claims description 21
- 238000002509 fluorescent in situ hybridization Methods 0.000 claims description 16
- 238000003556 assay Methods 0.000 claims description 15
- 230000002401 inhibitory effect Effects 0.000 claims description 15
- 229960004316 cisplatin Drugs 0.000 claims description 13
- DQLATGHUWYMOKM-UHFFFAOYSA-L cisplatin Chemical compound N[Pt](N)(Cl)Cl DQLATGHUWYMOKM-UHFFFAOYSA-L 0.000 claims description 13
- 239000000546 pharmaceutical excipient Substances 0.000 claims description 12
- 241000124008 Mammalia Species 0.000 claims description 11
- 238000012163 sequencing technique Methods 0.000 claims description 11
- 230000001404 mediated effect Effects 0.000 claims description 10
- 239000003937 drug carrier Substances 0.000 claims description 8
- 238000003745 diagnosis Methods 0.000 claims description 7
- 239000003085 diluting agent Substances 0.000 claims description 6
- 206010027476 Metastases Diseases 0.000 claims description 4
- 229940049920 malate Drugs 0.000 claims description 4
- 230000009401 metastasis Effects 0.000 claims description 4
- 230000004044 response Effects 0.000 claims description 4
- 230000001225 therapeutic effect Effects 0.000 claims description 4
- 230000036961 partial effect Effects 0.000 claims description 2
- 102100032218 Cytokine-inducible SH2-containing protein Human genes 0.000 claims 1
- 101000943420 Homo sapiens Cytokine-inducible SH2-containing protein Proteins 0.000 claims 1
- 230000001575 pathological effect Effects 0.000 claims 1
- 238000002976 reverse transcriptase assay Methods 0.000 claims 1
- 108020001507 fusion proteins Proteins 0.000 description 181
- 102000037865 fusion proteins Human genes 0.000 description 181
- 108091033319 polynucleotide Proteins 0.000 description 152
- 102000040430 polynucleotide Human genes 0.000 description 152
- 239000002157 polynucleotide Substances 0.000 description 152
- 239000000523 sample Substances 0.000 description 152
- 108090000623 proteins and genes Proteins 0.000 description 136
- 150000007523 nucleic acids Chemical class 0.000 description 79
- 108091006576 SLC34A2 Proteins 0.000 description 75
- 102100038437 Sodium-dependent phosphate transport protein 2B Human genes 0.000 description 75
- 210000004027 cell Anatomy 0.000 description 74
- 239000013615 primer Substances 0.000 description 70
- 102000039446 nucleic acids Human genes 0.000 description 69
- 108020004707 nucleic acids Proteins 0.000 description 69
- 239000000203 mixture Substances 0.000 description 55
- 101001082627 Homo sapiens HLA class II histocompatibility antigen gamma chain Proteins 0.000 description 53
- 102100030595 HLA class II histocompatibility antigen gamma chain Human genes 0.000 description 45
- 101001072499 Homo sapiens Golgi-associated PDZ and coiled-coil motif-containing protein Proteins 0.000 description 45
- 102100036675 Golgi-associated PDZ and coiled-coil motif-containing protein Human genes 0.000 description 44
- 239000000243 solution Substances 0.000 description 44
- 102000004169 proteins and genes Human genes 0.000 description 43
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 43
- WYURNTSHIVDZCO-UHFFFAOYSA-N Tetrahydrofuran Chemical compound C1CCOC1 WYURNTSHIVDZCO-UHFFFAOYSA-N 0.000 description 42
- 235000001014 amino acid Nutrition 0.000 description 42
- 229940024606 amino acid Drugs 0.000 description 39
- 230000002759 chromosomal effect Effects 0.000 description 39
- 230000000694 effects Effects 0.000 description 39
- 108700024394 Exon Proteins 0.000 description 38
- 150000001413 amino acids Chemical class 0.000 description 38
- 235000018102 proteins Nutrition 0.000 description 38
- 101150035397 Ros1 gene Proteins 0.000 description 37
- 239000002773 nucleotide Substances 0.000 description 37
- 125000003729 nucleotide group Chemical group 0.000 description 37
- 206010058467 Lung neoplasm malignant Diseases 0.000 description 36
- 108091000080 Phosphotransferase Proteins 0.000 description 36
- 208000020816 lung neoplasm Diseases 0.000 description 36
- 102000020233 phosphotransferase Human genes 0.000 description 36
- 201000005202 lung cancer Diseases 0.000 description 35
- 239000002585 base Substances 0.000 description 32
- 108020004414 DNA Proteins 0.000 description 31
- 239000003826 tablet Substances 0.000 description 31
- 239000003153 chemical reaction reagent Substances 0.000 description 30
- -1 L-malate salt Chemical class 0.000 description 26
- 230000000295 complement effect Effects 0.000 description 26
- 239000003112 inhibitor Substances 0.000 description 26
- ZWEHNKRNPOVVGH-UHFFFAOYSA-N 2-Butanone Chemical compound CCC(C)=O ZWEHNKRNPOVVGH-UHFFFAOYSA-N 0.000 description 25
- 238000009396 hybridization Methods 0.000 description 25
- 210000004899 c-terminal region Anatomy 0.000 description 24
- 239000000047 product Substances 0.000 description 24
- 230000035772 mutation Effects 0.000 description 23
- 238000002360 preparation method Methods 0.000 description 23
- 125000003275 alpha amino acid group Chemical group 0.000 description 21
- 230000005945 translocation Effects 0.000 description 21
- 239000000126 substance Substances 0.000 description 20
- 238000012360 testing method Methods 0.000 description 20
- 108091026890 Coding region Proteins 0.000 description 19
- 238000007901 in situ hybridization Methods 0.000 description 19
- 101150046784 Slc34a2 gene Proteins 0.000 description 18
- 238000006243 chemical reaction Methods 0.000 description 18
- 108020004999 messenger RNA Proteins 0.000 description 17
- IAZDPXIOMUYVGZ-UHFFFAOYSA-N DMSO Substances CS(C)=O IAZDPXIOMUYVGZ-UHFFFAOYSA-N 0.000 description 16
- 230000014509 gene expression Effects 0.000 description 16
- ZMXDDKWLCZADIW-UHFFFAOYSA-N N,N-Dimethylformamide Chemical compound CN(C)C=O ZMXDDKWLCZADIW-UHFFFAOYSA-N 0.000 description 15
- 210000000349 chromosome Anatomy 0.000 description 15
- 239000011541 reaction mixture Substances 0.000 description 15
- 239000007787 solid Substances 0.000 description 15
- YMWUJEATGCHHMB-UHFFFAOYSA-N Dichloromethane Chemical compound ClCCl YMWUJEATGCHHMB-UHFFFAOYSA-N 0.000 description 14
- IMNFDUFMRHMDMM-UHFFFAOYSA-N N-Heptane Chemical compound CCCCCCC IMNFDUFMRHMDMM-UHFFFAOYSA-N 0.000 description 14
- ONIQOQHATWINJY-UHFFFAOYSA-N cabozantinib Chemical compound C=12C=C(OC)C(OC)=CC2=NC=CC=1OC(C=C1)=CC=C1NC(=O)C1(C(=O)NC=2C=CC(F)=CC=2)CC1 ONIQOQHATWINJY-UHFFFAOYSA-N 0.000 description 14
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 13
- 210000003917 human chromosome Anatomy 0.000 description 13
- 210000001519 tissue Anatomy 0.000 description 13
- PEDCQBHIVMGVHV-UHFFFAOYSA-N Glycerine Chemical compound OCC(O)CO PEDCQBHIVMGVHV-UHFFFAOYSA-N 0.000 description 12
- 101100091501 Mus musculus Ros1 gene Proteins 0.000 description 12
- 230000002159 abnormal effect Effects 0.000 description 12
- JJWKPURADFRFRB-UHFFFAOYSA-N carbonyl sulfide Chemical compound O=C=S JJWKPURADFRFRB-UHFFFAOYSA-N 0.000 description 12
- 230000010261 cell growth Effects 0.000 description 12
- 238000005516 engineering process Methods 0.000 description 12
- 102000045479 human ROS1 Human genes 0.000 description 12
- BJEPYKJPYRNKOW-REOHCLBHSA-N (S)-malic acid Chemical compound OC(=O)[C@@H](O)CC(O)=O BJEPYKJPYRNKOW-REOHCLBHSA-N 0.000 description 10
- 101000579425 Homo sapiens Proto-oncogene tyrosine-protein kinase receptor Ret Proteins 0.000 description 10
- 230000003321 amplification Effects 0.000 description 10
- 230000000692 anti-sense effect Effects 0.000 description 10
- 239000002246 antineoplastic agent Substances 0.000 description 10
- 208000035475 disorder Diseases 0.000 description 10
- 238000003199 nucleic acid amplification method Methods 0.000 description 10
- VXEQRXJATQUJSN-UHFFFAOYSA-N 4-(6,7-dimethoxyquinolin-4-yl)oxyaniline Chemical compound C=12C=C(OC)C(OC)=CC2=NC=CC=1OC1=CC=C(N)C=C1 VXEQRXJATQUJSN-UHFFFAOYSA-N 0.000 description 9
- 239000002176 L01XE26 - Cabozantinib Substances 0.000 description 9
- OKKJLVBELUTLKV-UHFFFAOYSA-N Methanol Chemical compound OC OKKJLVBELUTLKV-UHFFFAOYSA-N 0.000 description 9
- DNIAPMSPPWPWGF-UHFFFAOYSA-N Propylene glycol Chemical compound CC(O)CO DNIAPMSPPWPWGF-UHFFFAOYSA-N 0.000 description 9
- 102100028286 Proto-oncogene tyrosine-protein kinase receptor Ret Human genes 0.000 description 9
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 9
- 238000012217 deletion Methods 0.000 description 9
- 230000037430 deletion Effects 0.000 description 9
- 125000005843 halogen group Chemical group 0.000 description 9
- 206010073071 hepatocellular carcinoma Diseases 0.000 description 9
- 238000004128 high performance liquid chromatography Methods 0.000 description 9
- 230000005764 inhibitory process Effects 0.000 description 9
- 229940043355 kinase inhibitor Drugs 0.000 description 9
- 239000003757 phosphotransferase inhibitor Substances 0.000 description 9
- 239000000725 suspension Substances 0.000 description 9
- 208000011580 syndromic disease Diseases 0.000 description 9
- IJGRMHOSHXDMSA-UHFFFAOYSA-N Atomic nitrogen Chemical compound N#N IJGRMHOSHXDMSA-UHFFFAOYSA-N 0.000 description 8
- 241000282412 Homo Species 0.000 description 8
- FXHOOIRPVKKKFG-UHFFFAOYSA-N N,N-Dimethylacetamide Chemical compound CN(C)C(C)=O FXHOOIRPVKKKFG-UHFFFAOYSA-N 0.000 description 8
- 108091034117 Oligonucleotide Proteins 0.000 description 8
- 238000004458 analytical method Methods 0.000 description 8
- 229940041181 antineoplastic drug Drugs 0.000 description 8
- 239000012472 biological sample Substances 0.000 description 8
- 239000000872 buffer Substances 0.000 description 8
- 239000002299 complementary DNA Substances 0.000 description 8
- 239000000284 extract Substances 0.000 description 8
- 238000001914 filtration Methods 0.000 description 8
- 239000001257 hydrogen Substances 0.000 description 8
- 229910052739 hydrogen Inorganic materials 0.000 description 8
- 238000002844 melting Methods 0.000 description 8
- 230000008018 melting Effects 0.000 description 8
- 238000001556 precipitation Methods 0.000 description 8
- 238000002560 therapeutic procedure Methods 0.000 description 8
- PFMAFXYUHZDKPY-UHFFFAOYSA-N 1-[(4-fluorophenyl)carbamoyl]cyclopropane-1-carboxylic acid Chemical compound C=1C=C(F)C=CC=1NC(=O)C1(C(=O)O)CC1 PFMAFXYUHZDKPY-UHFFFAOYSA-N 0.000 description 7
- UFHFLCQGNIYNRP-UHFFFAOYSA-N Hydrogen Chemical compound [H][H] UFHFLCQGNIYNRP-UHFFFAOYSA-N 0.000 description 7
- KFZMGEQAYNKOFK-UHFFFAOYSA-N Isopropanol Chemical compound CC(C)O KFZMGEQAYNKOFK-UHFFFAOYSA-N 0.000 description 7
- 238000012408 PCR amplification Methods 0.000 description 7
- 229940126394 ROS1 tyrosine kinase inhibitor Drugs 0.000 description 7
- 239000002253 acid Substances 0.000 description 7
- 229960001292 cabozantinib Drugs 0.000 description 7
- 238000003018 immunoassay Methods 0.000 description 7
- 239000007788 liquid Substances 0.000 description 7
- 239000007858 starting material Substances 0.000 description 7
- 238000013519 translation Methods 0.000 description 7
- 238000005292 vacuum distillation Methods 0.000 description 7
- WEVYAHXRMPXWCK-UHFFFAOYSA-N Acetonitrile Chemical compound CC#N WEVYAHXRMPXWCK-UHFFFAOYSA-N 0.000 description 6
- 206010069754 Acquired gene mutation Diseases 0.000 description 6
- OKTJSMMVPCPJKN-UHFFFAOYSA-N Carbon Chemical compound [C] OKTJSMMVPCPJKN-UHFFFAOYSA-N 0.000 description 6
- WSFSSNUMVMOOMR-UHFFFAOYSA-N Formaldehyde Chemical compound O=C WSFSSNUMVMOOMR-UHFFFAOYSA-N 0.000 description 6
- 102000004022 Protein-Tyrosine Kinases Human genes 0.000 description 6
- 108090000412 Protein-Tyrosine Kinases Proteins 0.000 description 6
- HEMHJVSKTPXQMS-UHFFFAOYSA-M Sodium hydroxide Chemical compound [OH-].[Na+] HEMHJVSKTPXQMS-UHFFFAOYSA-M 0.000 description 6
- DBMJMQXJHONAFJ-UHFFFAOYSA-M Sodium laurylsulphate Chemical compound [Na+].CCCCCCCCCCCCOS([O-])(=O)=O DBMJMQXJHONAFJ-UHFFFAOYSA-M 0.000 description 6
- ZMANZCXQSJIPKH-UHFFFAOYSA-N Triethylamine Chemical compound CCN(CC)CC ZMANZCXQSJIPKH-UHFFFAOYSA-N 0.000 description 6
- 230000004913 activation Effects 0.000 description 6
- 230000027455 binding Effects 0.000 description 6
- 238000001574 biopsy Methods 0.000 description 6
- 229910052799 carbon Inorganic materials 0.000 description 6
- 230000008711 chromosomal rearrangement Effects 0.000 description 6
- KRKNYBCHXYNGOX-UHFFFAOYSA-N citric acid Chemical compound OC(=O)CC(O)(C(O)=O)CC(O)=O KRKNYBCHXYNGOX-UHFFFAOYSA-N 0.000 description 6
- FDKLLWKMYAMLIF-UHFFFAOYSA-N cyclopropane-1,1-dicarboxylic acid Chemical compound OC(=O)C1(C(O)=O)CC1 FDKLLWKMYAMLIF-UHFFFAOYSA-N 0.000 description 6
- 229940079593 drug Drugs 0.000 description 6
- 239000003814 drug Substances 0.000 description 6
- 235000019441 ethanol Nutrition 0.000 description 6
- 230000002068 genetic effect Effects 0.000 description 6
- 210000004072 lung Anatomy 0.000 description 6
- 239000012528 membrane Substances 0.000 description 6
- 239000012074 organic phase Substances 0.000 description 6
- CTSLXHKWHWQRSH-UHFFFAOYSA-N oxalyl chloride Chemical compound ClC(=O)C(Cl)=O CTSLXHKWHWQRSH-UHFFFAOYSA-N 0.000 description 6
- 239000012071 phase Substances 0.000 description 6
- BWHMMNNQKKPAPP-UHFFFAOYSA-L potassium carbonate Chemical compound [K+].[K+].[O-]C([O-])=O BWHMMNNQKKPAPP-UHFFFAOYSA-L 0.000 description 6
- 235000019333 sodium laurylsulphate Nutrition 0.000 description 6
- 230000037439 somatic mutation Effects 0.000 description 6
- 238000006467 substitution reaction Methods 0.000 description 6
- 238000013518 transcription Methods 0.000 description 6
- 230000035897 transcription Effects 0.000 description 6
- JQWTVIFNRAALHM-UHFFFAOYSA-N 1-[(4-fluorophenyl)carbamoyl]cyclopropane-1-carbonyl chloride Chemical compound C1=CC(F)=CC=C1NC(=O)C1(C(Cl)=O)CC1 JQWTVIFNRAALHM-UHFFFAOYSA-N 0.000 description 5
- BHKKSKOHRFHHIN-MRVPVSSYSA-N 1-[[2-[(1R)-1-aminoethyl]-4-chlorophenyl]methyl]-2-sulfanylidene-5H-pyrrolo[3,2-d]pyrimidin-4-one Chemical compound N[C@H](C)C1=C(CN2C(NC(C3=C2C=CN3)=O)=S)C=CC(=C1)Cl BHKKSKOHRFHHIN-MRVPVSSYSA-N 0.000 description 5
- 108091023037 Aptamer Proteins 0.000 description 5
- 241001465754 Metazoa Species 0.000 description 5
- NGRZMRGYECEMTH-UHFFFAOYSA-N NC1=CC=C(F)C=C1.C=12C=C(OC)C(OC)=CC2=NC=CC=1OC1=CC=C(N)C=C1 Chemical compound NC1=CC=C(F)C=C1.C=12C=C(OC)C(OC)=CC2=NC=CC=1OC1=CC=C(N)C=C1 NGRZMRGYECEMTH-UHFFFAOYSA-N 0.000 description 5
- 108091028043 Nucleic acid sequence Proteins 0.000 description 5
- 102100037220 Syndecan-4 Human genes 0.000 description 5
- 208000009956 adenocarcinoma Diseases 0.000 description 5
- BJEPYKJPYRNKOW-UHFFFAOYSA-N alpha-hydroxysuccinic acid Natural products OC(=O)C(O)CC(O)=O BJEPYKJPYRNKOW-UHFFFAOYSA-N 0.000 description 5
- 230000037396 body weight Effects 0.000 description 5
- 239000003795 chemical substances by application Substances 0.000 description 5
- 125000004122 cyclic group Chemical group 0.000 description 5
- 239000012530 fluid Substances 0.000 description 5
- 229940125874 fusion protein inhibitor Drugs 0.000 description 5
- 125000004435 hydrogen atom Chemical group [H]* 0.000 description 5
- 239000004615 ingredient Substances 0.000 description 5
- 229910052740 iodine Inorganic materials 0.000 description 5
- 229940116298 l- malic acid Drugs 0.000 description 5
- 235000011090 malic acid Nutrition 0.000 description 5
- 239000000843 powder Substances 0.000 description 5
- 239000002987 primer (paints) Substances 0.000 description 5
- 208000016691 refractory malignant neoplasm Diseases 0.000 description 5
- 238000012216 screening Methods 0.000 description 5
- 239000011780 sodium chloride Substances 0.000 description 5
- 238000011144 upstream manufacturing Methods 0.000 description 5
- 238000001262 western blot Methods 0.000 description 5
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 4
- 0 *c(cc1)ccc1O Chemical compound *c(cc1)ccc1O 0.000 description 4
- VBICKXHEKHSIBG-UHFFFAOYSA-N 1-monostearoylglycerol Chemical compound CCCCCCCCCCCCCCCCCC(=O)OCC(O)CO VBICKXHEKHSIBG-UHFFFAOYSA-N 0.000 description 4
- PLIKAWJENQZMHA-UHFFFAOYSA-N 4-aminophenol Chemical compound NC1=CC=C(O)C=C1 PLIKAWJENQZMHA-UHFFFAOYSA-N 0.000 description 4
- WRVHQEYBCDPZEU-UHFFFAOYSA-N 4-chloro-6,7-dimethoxyquinoline Chemical compound C1=CC(Cl)=C2C=C(OC)C(OC)=CC2=N1 WRVHQEYBCDPZEU-UHFFFAOYSA-N 0.000 description 4
- WOVKYSAHUYNSMH-RRKCRQDMSA-N 5-bromodeoxyuridine Chemical compound C1[C@H](O)[C@@H](CO)O[C@H]1N1C(=O)NC(=O)C(Br)=C1 WOVKYSAHUYNSMH-RRKCRQDMSA-N 0.000 description 4
- 101710168331 ALK tyrosine kinase receptor Proteins 0.000 description 4
- 102100033793 ALK tyrosine kinase receptor Human genes 0.000 description 4
- 208000005443 Circulating Neoplastic Cells Diseases 0.000 description 4
- 230000004544 DNA amplification Effects 0.000 description 4
- 102000001301 EGF receptor Human genes 0.000 description 4
- 238000002965 ELISA Methods 0.000 description 4
- BZLVMXJERCGZMT-UHFFFAOYSA-N Methyl tert-butyl ether Chemical compound COC(C)(C)C BZLVMXJERCGZMT-UHFFFAOYSA-N 0.000 description 4
- 230000001594 aberrant effect Effects 0.000 description 4
- 239000004480 active ingredient Substances 0.000 description 4
- 150000001408 amides Chemical group 0.000 description 4
- 125000000539 amino acid group Chemical group 0.000 description 4
- 210000004369 blood Anatomy 0.000 description 4
- 239000008280 blood Substances 0.000 description 4
- 239000000969 carrier Substances 0.000 description 4
- 230000005754 cellular signaling Effects 0.000 description 4
- OSASVXMJTNOKOY-UHFFFAOYSA-N chlorobutanol Chemical compound CC(C)(O)C(Cl)(Cl)Cl OSASVXMJTNOKOY-UHFFFAOYSA-N 0.000 description 4
- 230000003247 decreasing effect Effects 0.000 description 4
- 230000018109 developmental process Effects 0.000 description 4
- 239000003995 emulsifying agent Substances 0.000 description 4
- 150000002148 esters Chemical class 0.000 description 4
- 101150016193 fig gene Proteins 0.000 description 4
- 235000011187 glycerol Nutrition 0.000 description 4
- 230000036541 health Effects 0.000 description 4
- 230000002055 immunohistochemical effect Effects 0.000 description 4
- 238000002372 labelling Methods 0.000 description 4
- 239000007937 lozenge Substances 0.000 description 4
- HQKMJHAJHXVSDF-UHFFFAOYSA-L magnesium stearate Chemical compound [Mg+2].CCCCCCCCCCCCCCCCCC([O-])=O.CCCCCCCCCCCCCCCCCC([O-])=O HQKMJHAJHXVSDF-UHFFFAOYSA-L 0.000 description 4
- 238000004519 manufacturing process Methods 0.000 description 4
- 238000013507 mapping Methods 0.000 description 4
- 239000002609 medium Substances 0.000 description 4
- 229910052757 nitrogen Inorganic materials 0.000 description 4
- 239000002245 particle Substances 0.000 description 4
- 229920001223 polyethylene glycol Polymers 0.000 description 4
- 230000002265 prevention Effects 0.000 description 4
- 230000008707 rearrangement Effects 0.000 description 4
- 230000002441 reversible effect Effects 0.000 description 4
- YGSDEFSMJLZEOE-UHFFFAOYSA-N salicylic acid Chemical compound OC(=O)C1=CC=CC=C1O YGSDEFSMJLZEOE-UHFFFAOYSA-N 0.000 description 4
- 230000035945 sensitivity Effects 0.000 description 4
- 239000002904 solvent Substances 0.000 description 4
- 239000000758 substrate Substances 0.000 description 4
- FYSNRJHAOHDILO-UHFFFAOYSA-N thionyl chloride Chemical compound ClS(Cl)=O FYSNRJHAOHDILO-UHFFFAOYSA-N 0.000 description 4
- 229940121358 tyrosine kinase inhibitor Drugs 0.000 description 4
- 239000005483 tyrosine kinase inhibitor Substances 0.000 description 4
- 239000013598 vector Substances 0.000 description 4
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 description 3
- QTBSBXVTEAMEQO-UHFFFAOYSA-N Acetic acid Chemical compound CC(O)=O QTBSBXVTEAMEQO-UHFFFAOYSA-N 0.000 description 3
- 108060006698 EGF receptor Proteins 0.000 description 3
- 102000004190 Enzymes Human genes 0.000 description 3
- 108090000790 Enzymes Proteins 0.000 description 3
- XEKOWRVHYACXOJ-UHFFFAOYSA-N Ethyl acetate Chemical compound CCOC(C)=O XEKOWRVHYACXOJ-UHFFFAOYSA-N 0.000 description 3
- 108010010803 Gelatin Proteins 0.000 description 3
- 208000034951 Genetic Translocation Diseases 0.000 description 3
- WHUUTDBJXJRKMK-UHFFFAOYSA-N Glutamic acid Natural products OC(=O)C(N)CCC(O)=O WHUUTDBJXJRKMK-UHFFFAOYSA-N 0.000 description 3
- 101000740519 Homo sapiens Syndecan-4 Proteins 0.000 description 3
- WHUUTDBJXJRKMK-VKHMYHEASA-N L-glutamic acid Chemical compound OC(=O)[C@@H](N)CCC(O)=O WHUUTDBJXJRKMK-VKHMYHEASA-N 0.000 description 3
- COLNVLDHVKWLRT-QMMMGPOBSA-N L-phenylalanine Chemical compound OC(=O)[C@@H](N)CC1=CC=CC=C1 COLNVLDHVKWLRT-QMMMGPOBSA-N 0.000 description 3
- MUBZPKHOEPUJKR-UHFFFAOYSA-N Oxalic acid Chemical compound OC(=O)C(O)=O MUBZPKHOEPUJKR-UHFFFAOYSA-N 0.000 description 3
- 229920001213 Polysorbate 20 Polymers 0.000 description 3
- OFOBLEOULBTSOW-UHFFFAOYSA-N Propanedioic acid Natural products OC(=O)CC(O)=O OFOBLEOULBTSOW-UHFFFAOYSA-N 0.000 description 3
- 108010029485 Protein Isoforms Proteins 0.000 description 3
- 102000001708 Protein Isoforms Human genes 0.000 description 3
- 102000001253 Protein Kinase Human genes 0.000 description 3
- 239000012980 RPMI-1640 medium Substances 0.000 description 3
- VYPSYNLAJGMNEJ-UHFFFAOYSA-N Silicium dioxide Chemical compound O=[Si]=O VYPSYNLAJGMNEJ-UHFFFAOYSA-N 0.000 description 3
- 206010041067 Small cell lung cancer Diseases 0.000 description 3
- 108020004459 Small interfering RNA Proteins 0.000 description 3
- 229920002472 Starch Polymers 0.000 description 3
- 102100033080 Tropomyosin alpha-3 chain Human genes 0.000 description 3
- 230000009471 action Effects 0.000 description 3
- 230000003213 activating effect Effects 0.000 description 3
- 239000000654 additive Substances 0.000 description 3
- 230000000996 additive effect Effects 0.000 description 3
- 239000002671 adjuvant Substances 0.000 description 3
- 125000000217 alkyl group Chemical group 0.000 description 3
- 125000004429 atom Chemical group 0.000 description 3
- 230000015572 biosynthetic process Effects 0.000 description 3
- 230000000903 blocking effect Effects 0.000 description 3
- 239000006172 buffering agent Substances 0.000 description 3
- HFCFMRYTXDINDK-WNQIDUERSA-N cabozantinib malate Chemical compound OC(=O)[C@@H](O)CC(O)=O.C=12C=C(OC)C(OC)=CC2=NC=CC=1OC(C=C1)=CC=C1NC(=O)C1(C(=O)NC=2C=CC(F)=CC=2)CC1 HFCFMRYTXDINDK-WNQIDUERSA-N 0.000 description 3
- 208000006990 cholangiocarcinoma Diseases 0.000 description 3
- 238000011161 development Methods 0.000 description 3
- 235000005911 diet Nutrition 0.000 description 3
- 230000037213 diet Effects 0.000 description 3
- 239000006185 dispersion Substances 0.000 description 3
- 239000007850 fluorescent dye Substances 0.000 description 3
- 239000008273 gelatin Substances 0.000 description 3
- 229920000159 gelatin Polymers 0.000 description 3
- 235000019322 gelatine Nutrition 0.000 description 3
- 235000011852 gelatine desserts Nutrition 0.000 description 3
- 208000005017 glioblastoma Diseases 0.000 description 3
- 208000014018 liver neoplasm Diseases 0.000 description 3
- 210000005265 lung cell Anatomy 0.000 description 3
- 239000006166 lysate Substances 0.000 description 3
- 230000001394 metastastic effect Effects 0.000 description 3
- 206010061289 metastatic neoplasm Diseases 0.000 description 3
- 238000010208 microarray analysis Methods 0.000 description 3
- 231100000590 oncogenic Toxicity 0.000 description 3
- 230000002246 oncogenic effect Effects 0.000 description 3
- 230000000144 pharmacologic effect Effects 0.000 description 3
- 235000010486 polyoxyethylene sorbitan monolaurate Nutrition 0.000 description 3
- 239000000256 polyoxyethylene sorbitan monolaurate Substances 0.000 description 3
- 229910000027 potassium carbonate Inorganic materials 0.000 description 3
- 230000003389 potentiating effect Effects 0.000 description 3
- 239000003755 preservative agent Substances 0.000 description 3
- 230000008569 process Effects 0.000 description 3
- 238000012545 processing Methods 0.000 description 3
- 239000000651 prodrug Substances 0.000 description 3
- 229940002612 prodrug Drugs 0.000 description 3
- 230000035755 proliferation Effects 0.000 description 3
- 229960004063 propylene glycol Drugs 0.000 description 3
- 108060006633 protein kinase Proteins 0.000 description 3
- 230000012743 protein tagging Effects 0.000 description 3
- 238000003127 radioimmunoassay Methods 0.000 description 3
- 238000003753 real-time PCR Methods 0.000 description 3
- 230000001105 regulatory effect Effects 0.000 description 3
- 238000003757 reverse transcription PCR Methods 0.000 description 3
- 210000002966 serum Anatomy 0.000 description 3
- 208000000587 small cell lung carcinoma Diseases 0.000 description 3
- 230000000392 somatic effect Effects 0.000 description 3
- 235000019698 starch Nutrition 0.000 description 3
- 239000008107 starch Substances 0.000 description 3
- 229940032147 starch Drugs 0.000 description 3
- 229960005322 streptomycin Drugs 0.000 description 3
- 239000000829 suppository Substances 0.000 description 3
- 239000000375 suspending agent Substances 0.000 description 3
- 208000024891 symptom Diseases 0.000 description 3
- 238000011285 therapeutic regimen Methods 0.000 description 3
- JOXIMZWYDAKGHI-UHFFFAOYSA-N toluene-4-sulfonic acid Chemical compound CC1=CC=C(S(O)(=O)=O)C=C1 JOXIMZWYDAKGHI-UHFFFAOYSA-N 0.000 description 3
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N (+)-Biotin Chemical compound N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 description 2
- PUPZLCDOIYMWBV-UHFFFAOYSA-N (+/-)-1,3-Butanediol Chemical compound CC(O)CCO PUPZLCDOIYMWBV-UHFFFAOYSA-N 0.000 description 2
- MTCFGRXMJLQNBG-REOHCLBHSA-N (2S)-2-Amino-3-hydroxypropansäure Chemical compound OC[C@H](N)C(O)=O MTCFGRXMJLQNBG-REOHCLBHSA-N 0.000 description 2
- JNYAEWCLZODPBN-JGWLITMVSA-N (2r,3r,4s)-2-[(1r)-1,2-dihydroxyethyl]oxolane-3,4-diol Chemical compound OC[C@@H](O)[C@H]1OC[C@H](O)[C@H]1O JNYAEWCLZODPBN-JGWLITMVSA-N 0.000 description 2
- 125000004169 (C1-C6) alkyl group Chemical group 0.000 description 2
- NGNBDVOYPDDBFK-UHFFFAOYSA-N 2-[2,4-di(pentan-2-yl)phenoxy]acetyl chloride Chemical compound CCCC(C)C1=CC=C(OCC(Cl)=O)C(C(C)CCC)=C1 NGNBDVOYPDDBFK-UHFFFAOYSA-N 0.000 description 2
- QKNYBSVHEMOAJP-UHFFFAOYSA-N 2-amino-2-(hydroxymethyl)propane-1,3-diol;hydron;chloride Chemical compound Cl.OCC(N)(CO)CO QKNYBSVHEMOAJP-UHFFFAOYSA-N 0.000 description 2
- XMIIGOLPHOKFCH-UHFFFAOYSA-N 3-phenylpropionic acid Chemical compound OC(=O)CCC1=CC=CC=C1 XMIIGOLPHOKFCH-UHFFFAOYSA-N 0.000 description 2
- FWBHETKCLVMNFS-UHFFFAOYSA-N 4',6-Diamino-2-phenylindol Chemical compound C1=CC(C(=N)N)=CC=C1C1=CC2=CC=C(C(N)=N)C=C2N1 FWBHETKCLVMNFS-UHFFFAOYSA-N 0.000 description 2
- VHYFNPMBLIVWCW-UHFFFAOYSA-N 4-Dimethylaminopyridine Chemical compound CN(C)C1=CC=NC=C1 VHYFNPMBLIVWCW-UHFFFAOYSA-N 0.000 description 2
- KRZCOLNOCZKSDF-UHFFFAOYSA-N 4-fluoroaniline Chemical compound NC1=CC=C(F)C=C1 KRZCOLNOCZKSDF-UHFFFAOYSA-N 0.000 description 2
- QOGPNCUTXVZQSL-UHFFFAOYSA-N 6,7-dimethoxy-1h-quinolin-4-one Chemical compound C1=CC(O)=C2C=C(OC)C(OC)=CC2=N1 QOGPNCUTXVZQSL-UHFFFAOYSA-N 0.000 description 2
- 229920001817 Agar Polymers 0.000 description 2
- 101000686029 Arabidopsis thaliana DNA glycosylase/AP lyase ROS1 Proteins 0.000 description 2
- CIWBSHSKHKDKBQ-JLAZNSOCSA-N Ascorbic acid Chemical compound OC[C@H](O)[C@H]1OC(=O)C(O)=C1O CIWBSHSKHKDKBQ-JLAZNSOCSA-N 0.000 description 2
- 241000416162 Astragalus gummifer Species 0.000 description 2
- 101100421200 Caenorhabditis elegans sep-1 gene Proteins 0.000 description 2
- VTYYLEPIZMXCLO-UHFFFAOYSA-L Calcium carbonate Chemical compound [Ca+2].[O-]C([O-])=O VTYYLEPIZMXCLO-UHFFFAOYSA-L 0.000 description 2
- 241000283707 Capra Species 0.000 description 2
- CURLTUGMZLYLDI-UHFFFAOYSA-N Carbon dioxide Chemical compound O=C=O CURLTUGMZLYLDI-UHFFFAOYSA-N 0.000 description 2
- 102000053642 Catalytic RNA Human genes 0.000 description 2
- 108090000994 Catalytic RNA Proteins 0.000 description 2
- VEXZGXHMUGYJMC-UHFFFAOYSA-M Chloride anion Chemical compound [Cl-] VEXZGXHMUGYJMC-UHFFFAOYSA-M 0.000 description 2
- RGHNJXZEOKUKBD-SQOUGZDYSA-N D-gluconic acid Chemical compound OC[C@@H](O)[C@@H](O)[C@H](O)[C@@H](O)C(O)=O RGHNJXZEOKUKBD-SQOUGZDYSA-N 0.000 description 2
- 238000000018 DNA microarray Methods 0.000 description 2
- 239000003155 DNA primer Substances 0.000 description 2
- ZHNUHDYFZUAESO-UHFFFAOYSA-N Formamide Chemical compound NC=O ZHNUHDYFZUAESO-UHFFFAOYSA-N 0.000 description 2
- VZCYOOQTPOCHFL-OWOJBTEDSA-N Fumaric acid Chemical compound OC(=O)\C=C\C(O)=O VZCYOOQTPOCHFL-OWOJBTEDSA-N 0.000 description 2
- 241000206672 Gelidium Species 0.000 description 2
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 2
- DHMQDGOQFOQNFH-UHFFFAOYSA-N Glycine Chemical compound NCC(O)=O DHMQDGOQFOQNFH-UHFFFAOYSA-N 0.000 description 2
- 108091093094 Glycol nucleic acid Proteins 0.000 description 2
- AEMRFAOFKBGASW-UHFFFAOYSA-N Glycolic acid Chemical compound OCC(O)=O AEMRFAOFKBGASW-UHFFFAOYSA-N 0.000 description 2
- 101001017855 Homo sapiens Leucine-rich repeats and immunoglobulin-like domains protein 3 Proteins 0.000 description 2
- 101001012157 Homo sapiens Receptor tyrosine-protein kinase erbB-2 Proteins 0.000 description 2
- 101100187408 Homo sapiens SLC34A2 gene Proteins 0.000 description 2
- 101000604039 Homo sapiens Sodium-dependent phosphate transport protein 2B Proteins 0.000 description 2
- 101000850794 Homo sapiens Tropomyosin alpha-3 chain Proteins 0.000 description 2
- 101000851007 Homo sapiens Vascular endothelial growth factor receptor 2 Proteins 0.000 description 2
- VEXZGXHMUGYJMC-UHFFFAOYSA-N Hydrochloric acid Chemical compound Cl VEXZGXHMUGYJMC-UHFFFAOYSA-N 0.000 description 2
- DGAQECJNVWCQMB-PUAWFVPOSA-M Ilexoside XXIX Chemical compound C[C@@H]1CC[C@@]2(CC[C@@]3(C(=CC[C@H]4[C@]3(CC[C@@H]5[C@@]4(CC[C@@H](C5(C)C)OS(=O)(=O)[O-])C)C)[C@@H]2[C@]1(C)O)C)C(=O)O[C@H]6[C@@H]([C@H]([C@@H]([C@H](O6)CO)O)O)O.[Na+] DGAQECJNVWCQMB-PUAWFVPOSA-M 0.000 description 2
- 206010062717 Increased upper airway secretion Diseases 0.000 description 2
- DCXYFEDJOCDNAF-REOHCLBHSA-N L-asparagine Chemical compound OC(=O)[C@@H](N)CC(N)=O DCXYFEDJOCDNAF-REOHCLBHSA-N 0.000 description 2
- CKLJMWTZIZZHCS-REOHCLBHSA-N L-aspartic acid Chemical compound OC(=O)[C@@H](N)CC(O)=O CKLJMWTZIZZHCS-REOHCLBHSA-N 0.000 description 2
- ZDXPYRJPNDTMRX-VKHMYHEASA-N L-glutamine Chemical compound OC(=O)[C@@H](N)CCC(N)=O ZDXPYRJPNDTMRX-VKHMYHEASA-N 0.000 description 2
- AGPKZVBTJJNPAG-WHFBIAKZSA-N L-isoleucine Chemical compound CC[C@H](C)[C@H](N)C(O)=O AGPKZVBTJJNPAG-WHFBIAKZSA-N 0.000 description 2
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 2
- KDXKERNSBIXSRK-YFKPBYRVSA-N L-lysine Chemical compound NCCCC[C@H](N)C(O)=O KDXKERNSBIXSRK-YFKPBYRVSA-N 0.000 description 2
- AYFVYJQAPQTCCC-GBXIJSLDSA-N L-threonine Chemical compound C[C@@H](O)[C@H](N)C(O)=O AYFVYJQAPQTCCC-GBXIJSLDSA-N 0.000 description 2
- OUYCCCASQSFEME-QMMMGPOBSA-N L-tyrosine Chemical compound OC(=O)[C@@H](N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-QMMMGPOBSA-N 0.000 description 2
- 102100033284 Leucine-rich repeats and immunoglobulin-like domains protein 3 Human genes 0.000 description 2
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 description 2
- AFVFQIVMOAPDHO-UHFFFAOYSA-N Methanesulfonic acid Chemical compound CS(O)(=O)=O AFVFQIVMOAPDHO-UHFFFAOYSA-N 0.000 description 2
- 238000000636 Northern blotting Methods 0.000 description 2
- 241000283973 Oryctolagus cuniculus Species 0.000 description 2
- 108091007960 PI3Ks Proteins 0.000 description 2
- KDLHZDBZIXYQEI-UHFFFAOYSA-N Palladium Chemical compound [Pd] KDLHZDBZIXYQEI-UHFFFAOYSA-N 0.000 description 2
- 102000003992 Peroxidases Human genes 0.000 description 2
- ISWSIDIOOBJBQZ-UHFFFAOYSA-N Phenol Chemical compound OC1=CC=CC=C1 ISWSIDIOOBJBQZ-UHFFFAOYSA-N 0.000 description 2
- 102000003993 Phosphatidylinositol 3-kinases Human genes 0.000 description 2
- 108090000430 Phosphatidylinositol 3-kinases Proteins 0.000 description 2
- NBIIXXVUZAFLBC-UHFFFAOYSA-N Phosphoric acid Chemical compound OP(O)(O)=O NBIIXXVUZAFLBC-UHFFFAOYSA-N 0.000 description 2
- 239000002202 Polyethylene glycol Substances 0.000 description 2
- 108010076504 Protein Sorting Signals Proteins 0.000 description 2
- LCTONWCANYUPML-UHFFFAOYSA-N Pyruvic acid Chemical compound CC(=O)C(O)=O LCTONWCANYUPML-UHFFFAOYSA-N 0.000 description 2
- 102100030086 Receptor tyrosine-protein kinase erbB-2 Human genes 0.000 description 2
- 208000006265 Renal cell carcinoma Diseases 0.000 description 2
- CDBYLPFSWZWCQE-UHFFFAOYSA-L Sodium Carbonate Chemical compound [Na+].[Na+].[O-]C([O-])=O CDBYLPFSWZWCQE-UHFFFAOYSA-L 0.000 description 2
- PXIPVTKHYLBLMZ-UHFFFAOYSA-N Sodium azide Chemical compound [Na+].[N-]=[N+]=[N-] PXIPVTKHYLBLMZ-UHFFFAOYSA-N 0.000 description 2
- 238000002105 Southern blotting Methods 0.000 description 2
- CZMRCDWAGMRECN-UGDNZRGBSA-N Sucrose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 CZMRCDWAGMRECN-UGDNZRGBSA-N 0.000 description 2
- 229930006000 Sucrose Natural products 0.000 description 2
- QAOWNCQODCNURD-UHFFFAOYSA-N Sulfuric acid Chemical compound OS(O)(=O)=O QAOWNCQODCNURD-UHFFFAOYSA-N 0.000 description 2
- 108010055215 Syndecan-4 Proteins 0.000 description 2
- 101710120037 Toxin CcdB Proteins 0.000 description 2
- 229920001615 Tragacanth Polymers 0.000 description 2
- DTQVDTLACAAQTR-UHFFFAOYSA-N Trifluoroacetic acid Chemical compound OC(=O)C(F)(F)F DTQVDTLACAAQTR-UHFFFAOYSA-N 0.000 description 2
- KZSNJWFQEVHDMF-UHFFFAOYSA-N Valine Chemical compound CC(C)C(N)C(O)=O KZSNJWFQEVHDMF-UHFFFAOYSA-N 0.000 description 2
- 102000005789 Vascular Endothelial Growth Factors Human genes 0.000 description 2
- 108010019530 Vascular Endothelial Growth Factors Proteins 0.000 description 2
- 102100033177 Vascular endothelial growth factor receptor 2 Human genes 0.000 description 2
- 238000010521 absorption reaction Methods 0.000 description 2
- 239000013543 active substance Substances 0.000 description 2
- 239000000443 aerosol Substances 0.000 description 2
- 235000010419 agar Nutrition 0.000 description 2
- 239000012298 atmosphere Substances 0.000 description 2
- 239000000440 bentonite Substances 0.000 description 2
- 235000012216 bentonite Nutrition 0.000 description 2
- 229910000278 bentonite Inorganic materials 0.000 description 2
- SVPXDRXYRYOSEX-UHFFFAOYSA-N bentoquatam Chemical compound O.O=[Si]=O.O=[Al]O[Al]=O SVPXDRXYRYOSEX-UHFFFAOYSA-N 0.000 description 2
- WPYMKLBDIGXBTP-UHFFFAOYSA-N benzoic acid Chemical compound OC(=O)C1=CC=CC=C1 WPYMKLBDIGXBTP-UHFFFAOYSA-N 0.000 description 2
- SESFRYSPDFLNCH-UHFFFAOYSA-N benzyl benzoate Chemical compound C=1C=CC=CC=1C(=O)OCC1=CC=CC=C1 SESFRYSPDFLNCH-UHFFFAOYSA-N 0.000 description 2
- WQZGKKKJIJFFOK-VFUOTHLCSA-N beta-D-glucose Chemical compound OC[C@H]1O[C@@H](O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-VFUOTHLCSA-N 0.000 description 2
- 239000011230 binding agent Substances 0.000 description 2
- 210000001124 body fluid Anatomy 0.000 description 2
- 239000010839 body fluid Substances 0.000 description 2
- 239000011575 calcium Substances 0.000 description 2
- 229910002091 carbon monoxide Inorganic materials 0.000 description 2
- 230000004663 cell proliferation Effects 0.000 description 2
- 235000010980 cellulose Nutrition 0.000 description 2
- 239000001913 cellulose Substances 0.000 description 2
- 229920002678 cellulose Polymers 0.000 description 2
- 238000005119 centrifugation Methods 0.000 description 2
- 229960004926 chlorobutanol Drugs 0.000 description 2
- 235000015165 citric acid Nutrition 0.000 description 2
- 238000000576 coating method Methods 0.000 description 2
- 229940034568 cometriq Drugs 0.000 description 2
- 238000012790 confirmation Methods 0.000 description 2
- 239000000994 contrast dye Substances 0.000 description 2
- 239000012043 crude product Substances 0.000 description 2
- 229940127089 cytotoxic agent Drugs 0.000 description 2
- 238000013461 design Methods 0.000 description 2
- 230000004069 differentiation Effects 0.000 description 2
- XBDQKXXYIPTUBI-UHFFFAOYSA-N dimethylselenoniopropionate Natural products CCC(O)=O XBDQKXXYIPTUBI-UHFFFAOYSA-N 0.000 description 2
- 238000006073 displacement reaction Methods 0.000 description 2
- 238000004090 dissolution Methods 0.000 description 2
- 238000004821 distillation Methods 0.000 description 2
- 239000000890 drug combination Substances 0.000 description 2
- 238000007877 drug screening Methods 0.000 description 2
- 239000000975 dye Substances 0.000 description 2
- 230000001804 emulsifying effect Effects 0.000 description 2
- 239000000839 emulsion Substances 0.000 description 2
- 230000029142 excretion Effects 0.000 description 2
- 239000013604 expression vector Substances 0.000 description 2
- 239000000706 filtrate Substances 0.000 description 2
- 238000009472 formulation Methods 0.000 description 2
- 235000013922 glutamic acid Nutrition 0.000 description 2
- 239000004220 glutamic acid Substances 0.000 description 2
- YQEMORVAKMFKLG-UHFFFAOYSA-N glycerine monostearate Natural products CCCCCCCCCCCCCCCCCC(=O)OC(CO)CO YQEMORVAKMFKLG-UHFFFAOYSA-N 0.000 description 2
- SVUQHVRAGMNPLW-UHFFFAOYSA-N glycerol monostearate Natural products CCCCCCCCCCCCCCCCC(=O)OCC(O)CO SVUQHVRAGMNPLW-UHFFFAOYSA-N 0.000 description 2
- 238000011532 immunohistochemical staining Methods 0.000 description 2
- 238000012744 immunostaining Methods 0.000 description 2
- 238000009169 immunotherapy Methods 0.000 description 2
- 238000000338 in vitro Methods 0.000 description 2
- 108010028930 invariant chain Proteins 0.000 description 2
- SUMDYPCJJOFFON-UHFFFAOYSA-N isethionic acid Chemical compound OCCS(O)(=O)=O SUMDYPCJJOFFON-UHFFFAOYSA-N 0.000 description 2
- JMMWKPVZQRWMSS-UHFFFAOYSA-N isopropanol acetate Natural products CC(C)OC(C)=O JMMWKPVZQRWMSS-UHFFFAOYSA-N 0.000 description 2
- 229940011051 isopropyl acetate Drugs 0.000 description 2
- GWYFCOCPABKNJV-UHFFFAOYSA-N isovaleric acid Chemical compound CC(C)CC(O)=O GWYFCOCPABKNJV-UHFFFAOYSA-N 0.000 description 2
- JVTAAEKCZFNVCJ-UHFFFAOYSA-N lactic acid Chemical compound CC(O)C(O)=O JVTAAEKCZFNVCJ-UHFFFAOYSA-N 0.000 description 2
- 230000003902 lesion Effects 0.000 description 2
- 235000019359 magnesium stearate Nutrition 0.000 description 2
- 239000000463 material Substances 0.000 description 2
- 239000011159 matrix material Substances 0.000 description 2
- 238000005259 measurement Methods 0.000 description 2
- 230000002503 metabolic effect Effects 0.000 description 2
- BDAGIHXWWSANSR-UHFFFAOYSA-N methanoic acid Natural products OC=O BDAGIHXWWSANSR-UHFFFAOYSA-N 0.000 description 2
- 238000002156 mixing Methods 0.000 description 2
- TXXHDPDFNKHHGW-UHFFFAOYSA-N muconic acid Chemical compound OC(=O)C=CC=CC(O)=O TXXHDPDFNKHHGW-UHFFFAOYSA-N 0.000 description 2
- SYSQUGFVNFXIIT-UHFFFAOYSA-N n-[4-(1,3-benzoxazol-2-yl)phenyl]-4-nitrobenzenesulfonamide Chemical class C1=CC([N+](=O)[O-])=CC=C1S(=O)(=O)NC1=CC=C(C=2OC3=CC=CC=C3N=2)C=C1 SYSQUGFVNFXIIT-UHFFFAOYSA-N 0.000 description 2
- FUZZWVXGSFPDMH-UHFFFAOYSA-N n-hexanoic acid Natural products CCCCCC(O)=O FUZZWVXGSFPDMH-UHFFFAOYSA-N 0.000 description 2
- OIPZNTLJVJGRCI-UHFFFAOYSA-M octadecanoyloxyaluminum;dihydrate Chemical compound O.O.CCCCCCCCCCCCCCCCCC(=O)O[Al] OIPZNTLJVJGRCI-UHFFFAOYSA-M 0.000 description 2
- 239000003921 oil Substances 0.000 description 2
- 235000019198 oils Nutrition 0.000 description 2
- 239000004006 olive oil Substances 0.000 description 2
- 235000008390 olive oil Nutrition 0.000 description 2
- 230000002018 overexpression Effects 0.000 description 2
- FJKROLUGYXJWQN-UHFFFAOYSA-N papa-hydroxy-benzoic acid Natural products OC(=O)C1=CC=C(O)C=C1 FJKROLUGYXJWQN-UHFFFAOYSA-N 0.000 description 2
- 108040007629 peroxidase activity proteins Proteins 0.000 description 2
- COLNVLDHVKWLRT-UHFFFAOYSA-N phenylalanine Natural products OC(=O)C(N)CC1=CC=CC=C1 COLNVLDHVKWLRT-UHFFFAOYSA-N 0.000 description 2
- YBYRMVIVWMBXKQ-UHFFFAOYSA-N phenylmethanesulfonyl fluoride Chemical compound FS(=O)(=O)CC1=CC=CC=C1 YBYRMVIVWMBXKQ-UHFFFAOYSA-N 0.000 description 2
- 208000026435 phlegm Diseases 0.000 description 2
- XHXFXVLFKHQFAL-UHFFFAOYSA-N phosphoryl trichloride Chemical compound ClP(Cl)(Cl)=O XHXFXVLFKHQFAL-UHFFFAOYSA-N 0.000 description 2
- 230000026731 phosphorylation Effects 0.000 description 2
- 238000006366 phosphorylation reaction Methods 0.000 description 2
- BASFCYQUMIYNBI-UHFFFAOYSA-N platinum Chemical compound [Pt] BASFCYQUMIYNBI-UHFFFAOYSA-N 0.000 description 2
- 230000000750 progressive effect Effects 0.000 description 2
- 230000002035 prolonged effect Effects 0.000 description 2
- 102000027426 receptor tyrosine kinases Human genes 0.000 description 2
- 108091008598 receptor tyrosine kinases Proteins 0.000 description 2
- 208000015347 renal cell adenocarcinoma Diseases 0.000 description 2
- 108091092562 ribozyme Proteins 0.000 description 2
- 125000006413 ring segment Chemical group 0.000 description 2
- 229960004889 salicylic acid Drugs 0.000 description 2
- 229910052709 silver Inorganic materials 0.000 description 2
- 239000004332 silver Substances 0.000 description 2
- 239000011734 sodium Substances 0.000 description 2
- 229910052708 sodium Inorganic materials 0.000 description 2
- MFRIHAYPQRLWNB-UHFFFAOYSA-N sodium tert-butoxide Chemical compound [Na+].CC(C)(C)[O-] MFRIHAYPQRLWNB-UHFFFAOYSA-N 0.000 description 2
- 239000012453 solvate Substances 0.000 description 2
- 238000000638 solvent extraction Methods 0.000 description 2
- 241000894007 species Species 0.000 description 2
- 239000003381 stabilizer Substances 0.000 description 2
- 125000001424 substituent group Chemical group 0.000 description 2
- 239000005720 sucrose Substances 0.000 description 2
- 239000004094 surface-active agent Substances 0.000 description 2
- 239000007916 tablet composition Substances 0.000 description 2
- YLQBMQCUIZJEEH-UHFFFAOYSA-N tetrahydrofuran Natural products C=1C=COC=1 YLQBMQCUIZJEEH-UHFFFAOYSA-N 0.000 description 2
- 235000010487 tragacanth Nutrition 0.000 description 2
- 239000000196 tragacanth Substances 0.000 description 2
- 229940116362 tragacanth Drugs 0.000 description 2
- VZCYOOQTPOCHFL-UHFFFAOYSA-N trans-butenedioic acid Natural products OC(=O)C=CC(O)=O VZCYOOQTPOCHFL-UHFFFAOYSA-N 0.000 description 2
- 102000027257 transmembrane receptors Human genes 0.000 description 2
- 108091008578 transmembrane receptors Proteins 0.000 description 2
- 239000003981 vehicle Substances 0.000 description 2
- 238000005406 washing Methods 0.000 description 2
- 239000001993 wax Substances 0.000 description 2
- 239000000080 wetting agent Substances 0.000 description 2
- 229940049068 xalkori Drugs 0.000 description 2
- QBYIENPQHBMVBV-HFEGYEGKSA-N (2R)-2-hydroxy-2-phenylacetic acid Chemical compound O[C@@H](C(O)=O)c1ccccc1.O[C@@H](C(O)=O)c1ccccc1 QBYIENPQHBMVBV-HFEGYEGKSA-N 0.000 description 1
- LNAZSHAWQACDHT-XIYTZBAFSA-N (2r,3r,4s,5r,6s)-4,5-dimethoxy-2-(methoxymethyl)-3-[(2s,3r,4s,5r,6r)-3,4,5-trimethoxy-6-(methoxymethyl)oxan-2-yl]oxy-6-[(2r,3r,4s,5r,6r)-4,5,6-trimethoxy-2-(methoxymethyl)oxan-3-yl]oxyoxane Chemical compound CO[C@@H]1[C@@H](OC)[C@H](OC)[C@@H](COC)O[C@H]1O[C@H]1[C@H](OC)[C@@H](OC)[C@H](O[C@H]2[C@@H]([C@@H](OC)[C@H](OC)O[C@@H]2COC)OC)O[C@@H]1COC LNAZSHAWQACDHT-XIYTZBAFSA-N 0.000 description 1
- BJEPYKJPYRNKOW-UWTATZPHSA-N (R)-malic acid Chemical class OC(=O)[C@H](O)CC(O)=O BJEPYKJPYRNKOW-UWTATZPHSA-N 0.000 description 1
- MIOPJNTWMNEORI-GMSGAONNSA-N (S)-camphorsulfonic acid Chemical compound C1C[C@@]2(CS(O)(=O)=O)C(=O)C[C@@H]1C2(C)C MIOPJNTWMNEORI-GMSGAONNSA-N 0.000 description 1
- ICLYJLBTOGPLMC-KVVVOXFISA-N (z)-octadec-9-enoate;tris(2-hydroxyethyl)azanium Chemical compound OCCN(CCO)CCO.CCCCCCCC\C=C/CCCCCCCC(O)=O ICLYJLBTOGPLMC-KVVVOXFISA-N 0.000 description 1
- WBYWAXJHAXSJNI-VOTSOKGWSA-M .beta-Phenylacrylic acid Natural products [O-]C(=O)\C=C\C1=CC=CC=C1 WBYWAXJHAXSJNI-VOTSOKGWSA-M 0.000 description 1
- 229940058015 1,3-butylene glycol Drugs 0.000 description 1
- QAPSNMNOIOSXSQ-YNEHKIRRSA-N 1-[(2r,4s,5r)-4-[tert-butyl(dimethyl)silyl]oxy-5-(hydroxymethyl)oxolan-2-yl]-5-methylpyrimidine-2,4-dione Chemical compound O=C1NC(=O)C(C)=CN1[C@@H]1O[C@H](CO)[C@@H](O[Si](C)(C)C(C)(C)C)C1 QAPSNMNOIOSXSQ-YNEHKIRRSA-N 0.000 description 1
- NDJNDUULNXNRQD-XKBRQERYSA-N 1-[(2r,4s,5s)-5-[bromo(hydroxy)methyl]-4-hydroxyoxolan-2-yl]pyrimidine-2,4-dione Chemical compound C1[C@H](O)[C@@H](C(Br)O)O[C@H]1N1C(=O)NC(=O)C=C1 NDJNDUULNXNRQD-XKBRQERYSA-N 0.000 description 1
- IXPNQXFRVYWDDI-UHFFFAOYSA-N 1-methyl-2,4-dioxo-1,3-diazinane-5-carboximidamide Chemical compound CN1CC(C(N)=N)C(=O)NC1=O IXPNQXFRVYWDDI-UHFFFAOYSA-N 0.000 description 1
- IIZPXYDJLKNOIY-JXPKJXOSSA-N 1-palmitoyl-2-arachidonoyl-sn-glycero-3-phosphocholine Chemical compound CCCCCCCCCCCCCCCC(=O)OC[C@H](COP([O-])(=O)OCC[N+](C)(C)C)OC(=O)CCC\C=C/C\C=C/C\C=C/C\C=C/CCCCC IIZPXYDJLKNOIY-JXPKJXOSSA-N 0.000 description 1
- 238000005160 1H NMR spectroscopy Methods 0.000 description 1
- GNENVASJJIUNER-UHFFFAOYSA-N 2,4,6-tricyclohexyloxy-1,3,5,2,4,6-trioxatriborinane Chemical compound C1CCCCC1OB1OB(OC2CCCCC2)OB(OC2CCCCC2)O1 GNENVASJJIUNER-UHFFFAOYSA-N 0.000 description 1
- OISVCGZHLKNMSJ-UHFFFAOYSA-N 2,6-dimethylpyridine Chemical compound CC1=CC=CC(C)=N1 OISVCGZHLKNMSJ-UHFFFAOYSA-N 0.000 description 1
- UPHOPMSGKZNELG-UHFFFAOYSA-N 2-hydroxynaphthalene-1-carboxylic acid Chemical compound C1=CC=C2C(C(=O)O)=C(O)C=CC2=C1 UPHOPMSGKZNELG-UHFFFAOYSA-N 0.000 description 1
- XLZYKTYMLBOINK-UHFFFAOYSA-N 3-(4-hydroxybenzoyl)benzoic acid Chemical compound OC(=O)C1=CC=CC(C(=O)C=2C=CC(O)=CC=2)=C1 XLZYKTYMLBOINK-UHFFFAOYSA-N 0.000 description 1
- BMYNFMYTOJXKLE-UHFFFAOYSA-N 3-azaniumyl-2-hydroxypropanoate Chemical compound NCC(O)C(O)=O BMYNFMYTOJXKLE-UHFFFAOYSA-N 0.000 description 1
- ZRPLANDPDWYOMZ-UHFFFAOYSA-N 3-cyclopentylpropionic acid Chemical compound OC(=O)CCC1CCCC1 ZRPLANDPDWYOMZ-UHFFFAOYSA-N 0.000 description 1
- OSWFIVFLDKOXQC-UHFFFAOYSA-N 4-(3-methoxyphenyl)aniline Chemical compound COC1=CC=CC(C=2C=CC(N)=CC=2)=C1 OSWFIVFLDKOXQC-UHFFFAOYSA-N 0.000 description 1
- RJWBTWIBUIGANW-UHFFFAOYSA-N 4-chlorobenzenesulfonic acid Chemical compound OS(=O)(=O)C1=CC=C(Cl)C=C1 RJWBTWIBUIGANW-UHFFFAOYSA-N 0.000 description 1
- BTJIUGUIPKRLHP-UHFFFAOYSA-M 4-nitrophenolate Chemical compound [O-]C1=CC=C([N+]([O-])=O)C=C1 BTJIUGUIPKRLHP-UHFFFAOYSA-M 0.000 description 1
- YRYKZXCOGVCXBN-UHFFFAOYSA-N 6,7-dimethoxy-4-(4-nitrophenoxy)quinoline Chemical compound C=12C=C(OC)C(OC)=CC2=NC=CC=1OC1=CC=C([N+]([O-])=O)C=C1 YRYKZXCOGVCXBN-UHFFFAOYSA-N 0.000 description 1
- IRECQXWRHWPICF-UHFFFAOYSA-N 6,7-dimethyl-4-(4-nitrophenoxy)quinoline Chemical compound C=12C=C(C)C(C)=CC2=NC=CC=1OC1=CC=C([N+]([O-])=O)C=C1 IRECQXWRHWPICF-UHFFFAOYSA-N 0.000 description 1
- ZCYVEMRRCGMTRW-UHFFFAOYSA-N 7553-56-2 Chemical group [I] ZCYVEMRRCGMTRW-UHFFFAOYSA-N 0.000 description 1
- HBAQYPYDRFILMT-UHFFFAOYSA-N 8-[3-(1-cyclopropylpyrazol-4-yl)-1H-pyrazolo[4,3-d]pyrimidin-5-yl]-3-methyl-3,8-diazabicyclo[3.2.1]octan-2-one Chemical class C1(CC1)N1N=CC(=C1)C1=NNC2=C1N=C(N=C2)N1C2C(N(CC1CC2)C)=O HBAQYPYDRFILMT-UHFFFAOYSA-N 0.000 description 1
- 244000215068 Acacia senegal Species 0.000 description 1
- 102000002260 Alkaline Phosphatase Human genes 0.000 description 1
- 108020004774 Alkaline Phosphatase Proteins 0.000 description 1
- GUBGYTABKSRVRQ-XLOQQCSPSA-N Alpha-Lactose Chemical compound O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@H]1O[C@@H]1[C@@H](CO)O[C@H](O)[C@H](O)[C@H]1O GUBGYTABKSRVRQ-XLOQQCSPSA-N 0.000 description 1
- 239000005995 Aluminium silicate Substances 0.000 description 1
- 108010039627 Aprotinin Proteins 0.000 description 1
- 229930091051 Arenine Natural products 0.000 description 1
- 239000004475 Arginine Substances 0.000 description 1
- DCXYFEDJOCDNAF-UHFFFAOYSA-N Asparagine Natural products OC(=O)C(N)CC(N)=O DCXYFEDJOCDNAF-UHFFFAOYSA-N 0.000 description 1
- 239000005711 Benzoic acid Substances 0.000 description 1
- 206010004593 Bile duct cancer Diseases 0.000 description 1
- WKBOTKDWSSQWDR-UHFFFAOYSA-N Bromine atom Chemical group [Br] WKBOTKDWSSQWDR-UHFFFAOYSA-N 0.000 description 1
- 239000004322 Butylated hydroxytoluene Substances 0.000 description 1
- NLZUEZXRPGMBCV-UHFFFAOYSA-N Butylhydroxytoluene Chemical compound CC1=CC(C(C)(C)C)=C(O)C(C(C)(C)C)=C1 NLZUEZXRPGMBCV-UHFFFAOYSA-N 0.000 description 1
- WWZKQHOCKIZLMA-UHFFFAOYSA-N Caprylic acid Natural products CCCCCCCC(O)=O WWZKQHOCKIZLMA-UHFFFAOYSA-N 0.000 description 1
- 229920002134 Carboxymethyl cellulose Polymers 0.000 description 1
- 241000282693 Cercopithecidae Species 0.000 description 1
- ZAMOUSCENKQFHK-UHFFFAOYSA-N Chlorine atom Chemical group [Cl] ZAMOUSCENKQFHK-UHFFFAOYSA-N 0.000 description 1
- WBYWAXJHAXSJNI-SREVYHEPSA-N Cinnamic acid Chemical compound OC(=O)\C=C/C1=CC=CC=C1 WBYWAXJHAXSJNI-SREVYHEPSA-N 0.000 description 1
- 229920002785 Croscarmellose sodium Polymers 0.000 description 1
- LVZWSLJZHVFIQJ-UHFFFAOYSA-N Cyclopropane Chemical compound C1CC1 LVZWSLJZHVFIQJ-UHFFFAOYSA-N 0.000 description 1
- FBPFZTCFMRRESA-FSIIMWSLSA-N D-Glucitol Natural products OC[C@H](O)[C@H](O)[C@@H](O)[C@H](O)CO FBPFZTCFMRRESA-FSIIMWSLSA-N 0.000 description 1
- FBPFZTCFMRRESA-KVTDHHQDSA-N D-Mannitol Chemical compound OC[C@@H](O)[C@@H](O)[C@H](O)[C@H](O)CO FBPFZTCFMRRESA-KVTDHHQDSA-N 0.000 description 1
- FBPFZTCFMRRESA-JGWLITMVSA-N D-glucitol Chemical compound OC[C@H](O)[C@@H](O)[C@H](O)[C@H](O)CO FBPFZTCFMRRESA-JGWLITMVSA-N 0.000 description 1
- RGHNJXZEOKUKBD-UHFFFAOYSA-N D-gluconic acid Natural products OCC(O)C(O)C(O)C(O)C(O)=O RGHNJXZEOKUKBD-UHFFFAOYSA-N 0.000 description 1
- 108020001019 DNA Primers Proteins 0.000 description 1
- 108020003215 DNA Probes Proteins 0.000 description 1
- 238000007400 DNA extraction Methods 0.000 description 1
- 239000003298 DNA probe Substances 0.000 description 1
- 238000001712 DNA sequencing Methods 0.000 description 1
- 241001269238 Data Species 0.000 description 1
- FEWJPZIEWOKRBE-JCYAYHJZSA-N Dextrotartaric acid Chemical compound OC(=O)[C@H](O)[C@@H](O)C(O)=O FEWJPZIEWOKRBE-JCYAYHJZSA-N 0.000 description 1
- 235000019739 Dicalciumphosphate Nutrition 0.000 description 1
- 206010013710 Drug interaction Diseases 0.000 description 1
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 1
- LVGKNOAMLMIIKO-UHFFFAOYSA-N Elaidinsaeure-aethylester Natural products CCCCCCCCC=CCCCCCCCC(=O)OCC LVGKNOAMLMIIKO-UHFFFAOYSA-N 0.000 description 1
- OTMSDBZUPAUEDD-UHFFFAOYSA-N Ethane Chemical compound CC OTMSDBZUPAUEDD-UHFFFAOYSA-N 0.000 description 1
- 239000005977 Ethylene Substances 0.000 description 1
- 102100020903 Ezrin Human genes 0.000 description 1
- 108091008794 FGF receptors Proteins 0.000 description 1
- 102000002090 Fibronectin type III Human genes 0.000 description 1
- 108050009401 Fibronectin type III Proteins 0.000 description 1
- 239000004471 Glycine Substances 0.000 description 1
- 102000003886 Glycoproteins Human genes 0.000 description 1
- 108090000288 Glycoproteins Proteins 0.000 description 1
- 229920000084 Gum arabic Polymers 0.000 description 1
- 101710119866 HLA class II histocompatibility antigen gamma chain Proteins 0.000 description 1
- 102000006354 HLA-DR Antigens Human genes 0.000 description 1
- 108010058597 HLA-DR Antigens Proteins 0.000 description 1
- 101000854648 Homo sapiens Ezrin Proteins 0.000 description 1
- 108010001336 Horseradish Peroxidase Proteins 0.000 description 1
- 102000003839 Human Proteins Human genes 0.000 description 1
- 108090000144 Human Proteins Proteins 0.000 description 1
- 108060003951 Immunoglobulin Proteins 0.000 description 1
- 208000026350 Inborn Genetic disease Diseases 0.000 description 1
- 101150023321 KIF5B gene Proteins 0.000 description 1
- 102000010638 Kinesin Human genes 0.000 description 1
- 108010063296 Kinesin Proteins 0.000 description 1
- QNAYBMKLOCPYGJ-REOHCLBHSA-N L-alanine Chemical compound C[C@H](N)C(O)=O QNAYBMKLOCPYGJ-REOHCLBHSA-N 0.000 description 1
- 150000008575 L-amino acids Chemical class 0.000 description 1
- ODKSFYDXXFIFQN-BYPYZUCNSA-P L-argininium(2+) Chemical compound NC(=[NH2+])NCCC[C@H]([NH3+])C(O)=O ODKSFYDXXFIFQN-BYPYZUCNSA-P 0.000 description 1
- HNDVDQJCIGZPNO-YFKPBYRVSA-N L-histidine Chemical compound OC(=O)[C@@H](N)CC1=CN=CN1 HNDVDQJCIGZPNO-YFKPBYRVSA-N 0.000 description 1
- FFEARJCKVFRZRR-BYPYZUCNSA-N L-methionine Chemical compound CSCC[C@H](N)C(O)=O FFEARJCKVFRZRR-BYPYZUCNSA-N 0.000 description 1
- QIVBCDIJIAJPQS-VIFPVBQESA-N L-tryptophane Chemical compound C1=CC=C2C(C[C@H](N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-VIFPVBQESA-N 0.000 description 1
- KZSNJWFQEVHDMF-BYPYZUCNSA-N L-valine Chemical compound CC(C)[C@H](N)C(O)=O KZSNJWFQEVHDMF-BYPYZUCNSA-N 0.000 description 1
- 239000005411 L01XE02 - Gefitinib Substances 0.000 description 1
- 239000005551 L01XE03 - Erlotinib Substances 0.000 description 1
- 238000007397 LAMP assay Methods 0.000 description 1
- GUBGYTABKSRVRQ-QKKXKWKRSA-N Lactose Natural products OC[C@H]1O[C@@H](O[C@H]2[C@H](O)[C@@H](O)C(O)O[C@@H]2CO)[C@H](O)[C@@H](O)[C@H]1O GUBGYTABKSRVRQ-QKKXKWKRSA-N 0.000 description 1
- 240000007472 Leucaena leucocephala Species 0.000 description 1
- 235000010643 Leucaena leucocephala Nutrition 0.000 description 1
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 description 1
- GDBQQVLCIARPGH-UHFFFAOYSA-N Leupeptin Natural products CC(C)CC(NC(C)=O)C(=O)NC(CC(C)C)C(=O)NC(C=O)CCCN=C(N)N GDBQQVLCIARPGH-UHFFFAOYSA-N 0.000 description 1
- 102000003960 Ligases Human genes 0.000 description 1
- 108090000364 Ligases Proteins 0.000 description 1
- 108090001030 Lipoproteins Proteins 0.000 description 1
- 102000004895 Lipoproteins Human genes 0.000 description 1
- 239000004472 Lysine Substances 0.000 description 1
- 240000003183 Manihot esculenta Species 0.000 description 1
- 235000016735 Manihot esculenta subsp esculenta Nutrition 0.000 description 1
- 229930195725 Mannitol Natural products 0.000 description 1
- 102000018697 Membrane Proteins Human genes 0.000 description 1
- 108010052285 Membrane Proteins Proteins 0.000 description 1
- 229920000168 Microcrystalline cellulose Polymers 0.000 description 1
- TXXHDPDFNKHHGW-CCAGOZQPSA-N Muconic acid Natural products OC(=O)\C=C/C=C\C(O)=O TXXHDPDFNKHHGW-CCAGOZQPSA-N 0.000 description 1
- 241001529936 Murinae Species 0.000 description 1
- 108010021466 Mutant Proteins Proteins 0.000 description 1
- 102000008300 Mutant Proteins Human genes 0.000 description 1
- 125000000729 N-terminal amino-acid group Chemical group 0.000 description 1
- 229910020700 Na3VO4 Inorganic materials 0.000 description 1
- GRYLNZFGIOXLOG-UHFFFAOYSA-N Nitric acid Chemical compound O[N+]([O-])=O GRYLNZFGIOXLOG-UHFFFAOYSA-N 0.000 description 1
- 108091005461 Nucleic proteins Proteins 0.000 description 1
- 108010038807 Oligopeptides Proteins 0.000 description 1
- 102000015636 Oligopeptides Human genes 0.000 description 1
- 206010033128 Ovarian cancer Diseases 0.000 description 1
- 206010061535 Ovarian neoplasm Diseases 0.000 description 1
- 229910019142 PO4 Inorganic materials 0.000 description 1
- 235000019483 Peanut oil Nutrition 0.000 description 1
- 108091093037 Peptide nucleic acid Proteins 0.000 description 1
- 108010092528 Phosphate Transport Proteins Proteins 0.000 description 1
- 102000016462 Phosphate Transport Proteins Human genes 0.000 description 1
- 208000002151 Pleural effusion Diseases 0.000 description 1
- 229920003171 Poly (ethylene oxide) Polymers 0.000 description 1
- 239000004952 Polyamide Substances 0.000 description 1
- 239000004698 Polyethylene Substances 0.000 description 1
- 241000288906 Primates Species 0.000 description 1
- 206010036790 Productive cough Diseases 0.000 description 1
- 201000003454 Pulmonary alveolar microlithiasis Diseases 0.000 description 1
- IWYDHOAUDWTVEP-UHFFFAOYSA-N R-2-phenyl-2-hydroxyacetic acid Natural products OC(=O)C(O)C1=CC=CC=C1 IWYDHOAUDWTVEP-UHFFFAOYSA-N 0.000 description 1
- 108091030071 RNAI Proteins 0.000 description 1
- 108090000873 Receptor Protein-Tyrosine Kinases Proteins 0.000 description 1
- 108020004511 Recombinant DNA Proteins 0.000 description 1
- 108700005075 Regulator Genes Proteins 0.000 description 1
- 241000283984 Rodentia Species 0.000 description 1
- YASAKCUCGLMORW-UHFFFAOYSA-N Rosiglitazone Chemical compound C=1C=CC=NC=1N(C)CCOC(C=C1)=CC=C1CC1SC(=O)NC1=O YASAKCUCGLMORW-UHFFFAOYSA-N 0.000 description 1
- 108010017324 STAT3 Transcription Factor Proteins 0.000 description 1
- MTCFGRXMJLQNBG-UHFFFAOYSA-N Serine Natural products OCC(N)C(O)=O MTCFGRXMJLQNBG-UHFFFAOYSA-N 0.000 description 1
- 102100024040 Signal transducer and activator of transcription 3 Human genes 0.000 description 1
- 108091027967 Small hairpin RNA Proteins 0.000 description 1
- 102000013996 Sodium-dependent phosphate transport protein 2B Human genes 0.000 description 1
- 108050003877 Sodium-dependent phosphate transport protein 2B Proteins 0.000 description 1
- 235000002595 Solanum tuberosum Nutrition 0.000 description 1
- 244000061456 Solanum tuberosum Species 0.000 description 1
- 235000021355 Stearic acid Nutrition 0.000 description 1
- SSZBUIDZHHWXNJ-UHFFFAOYSA-N Stearinsaeure-hexadecylester Natural products CCCCCCCCCCCCCCCCCC(=O)OCCCCCCCCCCCCCCCC SSZBUIDZHHWXNJ-UHFFFAOYSA-N 0.000 description 1
- KDYFGRWQOYBRFD-UHFFFAOYSA-N Succinic acid Natural products OC(=O)CCC(O)=O KDYFGRWQOYBRFD-UHFFFAOYSA-N 0.000 description 1
- 102220600990 Syndecan-4_E34R_mutation Human genes 0.000 description 1
- FEWJPZIEWOKRBE-UHFFFAOYSA-N Tartaric acid Natural products [H+].[H+].[O-]C(=O)C(O)C(O)C([O-])=O FEWJPZIEWOKRBE-UHFFFAOYSA-N 0.000 description 1
- 244000299461 Theobroma cacao Species 0.000 description 1
- 235000009470 Theobroma cacao Nutrition 0.000 description 1
- AYFVYJQAPQTCCC-UHFFFAOYSA-N Threonine Natural products CC(O)C(N)C(O)=O AYFVYJQAPQTCCC-UHFFFAOYSA-N 0.000 description 1
- 239000004473 Threonine Substances 0.000 description 1
- 208000024770 Thyroid neoplasm Diseases 0.000 description 1
- 101710091952 Tropomyosin alpha-3 chain Proteins 0.000 description 1
- QIVBCDIJIAJPQS-UHFFFAOYSA-N Tryptophan Natural products C1=CC=C2C(CC(N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-UHFFFAOYSA-N 0.000 description 1
- 229940091171 VEGFR-2 tyrosine kinase inhibitor Drugs 0.000 description 1
- 240000008042 Zea mays Species 0.000 description 1
- 235000005824 Zea mays ssp. parviglumis Nutrition 0.000 description 1
- 235000002017 Zea mays subsp mays Nutrition 0.000 description 1
- 239000001089 [(2R)-oxolan-2-yl]methanol Substances 0.000 description 1
- CQODGVQBRIGKLJ-UHFFFAOYSA-L [Na+].[Na+].[O-]OOO[O-] Chemical compound [Na+].[Na+].[O-]OOO[O-] CQODGVQBRIGKLJ-UHFFFAOYSA-L 0.000 description 1
- 239000002250 absorbent Substances 0.000 description 1
- 230000002745 absorbent Effects 0.000 description 1
- 235000010489 acacia gum Nutrition 0.000 description 1
- 239000000205 acacia gum Substances 0.000 description 1
- 150000001242 acetic acid derivatives Chemical class 0.000 description 1
- DPXJVFZANSGRMM-UHFFFAOYSA-N acetic acid;2,3,4,5,6-pentahydroxyhexanal;sodium Chemical compound [Na].CC(O)=O.OCC(O)C(O)C(O)C(O)C=O DPXJVFZANSGRMM-UHFFFAOYSA-N 0.000 description 1
- 150000007513 acids Chemical class 0.000 description 1
- 239000011149 active material Substances 0.000 description 1
- 239000008186 active pharmaceutical agent Substances 0.000 description 1
- 230000001464 adherent effect Effects 0.000 description 1
- 238000000246 agarose gel electrophoresis Methods 0.000 description 1
- 235000004279 alanine Nutrition 0.000 description 1
- 150000001298 alcohols Chemical class 0.000 description 1
- 235000010443 alginic acid Nutrition 0.000 description 1
- 239000000783 alginic acid Substances 0.000 description 1
- 229920000615 alginic acid Polymers 0.000 description 1
- 229960001126 alginic acid Drugs 0.000 description 1
- 150000004781 alginic acids Chemical class 0.000 description 1
- 125000005907 alkyl ester group Chemical group 0.000 description 1
- 150000001371 alpha-amino acids Chemical class 0.000 description 1
- 235000008206 alpha-amino acids Nutrition 0.000 description 1
- 230000004075 alteration Effects 0.000 description 1
- 229910052782 aluminium Inorganic materials 0.000 description 1
- XAGFODPZIPBFFR-UHFFFAOYSA-N aluminium Chemical compound [Al] XAGFODPZIPBFFR-UHFFFAOYSA-N 0.000 description 1
- 229910000147 aluminium phosphate Inorganic materials 0.000 description 1
- 235000012211 aluminium silicate Nutrition 0.000 description 1
- 230000033115 angiogenesis Effects 0.000 description 1
- 239000003242 anti bacterial agent Substances 0.000 description 1
- 230000000844 anti-bacterial effect Effects 0.000 description 1
- 230000002155 anti-virotic effect Effects 0.000 description 1
- 229940121375 antifungal agent Drugs 0.000 description 1
- 239000003429 antifungal agent Substances 0.000 description 1
- 239000003963 antioxidant agent Substances 0.000 description 1
- 235000006708 antioxidants Nutrition 0.000 description 1
- 229940027983 antiseptic and disinfectant quaternary ammonium compound Drugs 0.000 description 1
- 229960004405 aprotinin Drugs 0.000 description 1
- 239000008346 aqueous phase Substances 0.000 description 1
- 239000007864 aqueous solution Substances 0.000 description 1
- 239000008135 aqueous vehicle Substances 0.000 description 1
- ODKSFYDXXFIFQN-UHFFFAOYSA-N arginine Natural products OC(=O)C(N)CCCNC(N)=N ODKSFYDXXFIFQN-UHFFFAOYSA-N 0.000 description 1
- 125000003710 aryl alkyl group Chemical group 0.000 description 1
- 125000003118 aryl group Chemical group 0.000 description 1
- 235000010323 ascorbic acid Nutrition 0.000 description 1
- 229960005070 ascorbic acid Drugs 0.000 description 1
- 239000011668 ascorbic acid Substances 0.000 description 1
- 235000009582 asparagine Nutrition 0.000 description 1
- 229960001230 asparagine Drugs 0.000 description 1
- 235000003704 aspartic acid Nutrition 0.000 description 1
- 238000013475 authorization Methods 0.000 description 1
- 230000035578 autophosphorylation Effects 0.000 description 1
- 229960000686 benzalkonium chloride Drugs 0.000 description 1
- SRSXLGNVWSONIS-UHFFFAOYSA-N benzenesulfonic acid Chemical compound OS(=O)(=O)C1=CC=CC=C1 SRSXLGNVWSONIS-UHFFFAOYSA-N 0.000 description 1
- 229940092714 benzenesulfonic acid Drugs 0.000 description 1
- 235000010233 benzoic acid Nutrition 0.000 description 1
- 229960002903 benzyl benzoate Drugs 0.000 description 1
- 125000001797 benzyl group Chemical group [H]C1=C([H])C([H])=C(C([H])=C1[H])C([H])([H])* 0.000 description 1
- CADWTSSKOVRVJC-UHFFFAOYSA-N benzyl(dimethyl)azanium;chloride Chemical compound [Cl-].C[NH+](C)CC1=CC=CC=C1 CADWTSSKOVRVJC-UHFFFAOYSA-N 0.000 description 1
- GONOPSZTUGRENK-UHFFFAOYSA-N benzyl(trichloro)silane Chemical compound Cl[Si](Cl)(Cl)CC1=CC=CC=C1 GONOPSZTUGRENK-UHFFFAOYSA-N 0.000 description 1
- OQFSQFPPLPISGP-UHFFFAOYSA-N beta-carboxyaspartic acid Natural products OC(=O)C(N)C(C(O)=O)C(O)=O OQFSQFPPLPISGP-UHFFFAOYSA-N 0.000 description 1
- 208000026900 bile duct neoplasm Diseases 0.000 description 1
- 229960002685 biotin Drugs 0.000 description 1
- 235000020958 biotin Nutrition 0.000 description 1
- 239000011616 biotin Substances 0.000 description 1
- OWMVSZAMULFTJU-UHFFFAOYSA-N bis-tris Chemical compound OCCN(CCO)C(CO)(CO)CO OWMVSZAMULFTJU-UHFFFAOYSA-N 0.000 description 1
- 230000036760 body temperature Effects 0.000 description 1
- 239000012267 brine Substances 0.000 description 1
- GDTBXPJZTBHREO-UHFFFAOYSA-N bromine Chemical group BrBr GDTBXPJZTBHREO-UHFFFAOYSA-N 0.000 description 1
- 229910052794 bromium Inorganic materials 0.000 description 1
- 239000007853 buffer solution Substances 0.000 description 1
- 235000019437 butane-1,3-diol Nutrition 0.000 description 1
- KDYFGRWQOYBRFD-NUQCWPJISA-N butanedioic acid Chemical compound O[14C](=O)CC[14C](O)=O KDYFGRWQOYBRFD-NUQCWPJISA-N 0.000 description 1
- 235000014121 butter Nutrition 0.000 description 1
- 235000010354 butylated hydroxytoluene Nutrition 0.000 description 1
- 229940095259 butylated hydroxytoluene Drugs 0.000 description 1
- 238000010805 cDNA synthesis kit Methods 0.000 description 1
- 229910000019 calcium carbonate Inorganic materials 0.000 description 1
- 235000010216 calcium carbonate Nutrition 0.000 description 1
- 239000001506 calcium phosphate Substances 0.000 description 1
- CJZGTCYPCWQAJB-UHFFFAOYSA-L calcium stearate Chemical compound [Ca+2].CCCCCCCCCCCCCCCCCC([O-])=O.CCCCCCCCCCCCCCCCCC([O-])=O CJZGTCYPCWQAJB-UHFFFAOYSA-L 0.000 description 1
- 235000013539 calcium stearate Nutrition 0.000 description 1
- 239000008116 calcium stearate Substances 0.000 description 1
- 230000001914 calming effect Effects 0.000 description 1
- 239000001569 carbon dioxide Substances 0.000 description 1
- 229910002092 carbon dioxide Inorganic materials 0.000 description 1
- 235000010948 carboxy methyl cellulose Nutrition 0.000 description 1
- 239000001768 carboxy methyl cellulose Substances 0.000 description 1
- 150000001732 carboxylic acid derivatives Chemical group 0.000 description 1
- 239000008112 carboxymethyl-cellulose Substances 0.000 description 1
- 231100000504 carcinogenesis Toxicity 0.000 description 1
- 239000004359 castor oil Substances 0.000 description 1
- 235000019438 castor oil Nutrition 0.000 description 1
- 229960000541 cetyl alcohol Drugs 0.000 description 1
- 238000012512 characterization method Methods 0.000 description 1
- 239000003638 chemical reducing agent Substances 0.000 description 1
- 239000000460 chlorine Chemical group 0.000 description 1
- 229910052801 chlorine Inorganic materials 0.000 description 1
- 238000003200 chromosome mapping Methods 0.000 description 1
- 235000013985 cinnamic acid Nutrition 0.000 description 1
- 229930016911 cinnamic acid Natural products 0.000 description 1
- 150000001860 citric acid derivatives Chemical class 0.000 description 1
- 238000003776 cleavage reaction Methods 0.000 description 1
- 238000010367 cloning Methods 0.000 description 1
- 239000011248 coating agent Substances 0.000 description 1
- 230000000052 comparative effect Effects 0.000 description 1
- 230000006552 constitutive activation Effects 0.000 description 1
- 239000012059 conventional drug carrier Substances 0.000 description 1
- 235000005822 corn Nutrition 0.000 description 1
- 235000012343 cottonseed oil Nutrition 0.000 description 1
- 239000002385 cottonseed oil Substances 0.000 description 1
- 229960001681 croscarmellose sodium Drugs 0.000 description 1
- 235000010947 crosslinked sodium carboxy methyl cellulose Nutrition 0.000 description 1
- 125000000753 cycloalkyl group Chemical group 0.000 description 1
- FEHLGOYZDFFMND-UHFFFAOYSA-N cyclopropane-1,1-dicarboxamide Chemical compound NC(=O)C1(C(N)=O)CC1 FEHLGOYZDFFMND-UHFFFAOYSA-N 0.000 description 1
- 230000007423 decrease Effects 0.000 description 1
- 230000007547 defect Effects 0.000 description 1
- 230000003111 delayed effect Effects 0.000 description 1
- 230000002939 deleterious effect Effects 0.000 description 1
- 229940124447 delivery agent Drugs 0.000 description 1
- KXGVEGMKQFWNSR-LLQZFEROSA-N deoxycholic acid Chemical compound C([C@H]1CC2)[C@H](O)CC[C@]1(C)[C@@H]1[C@@H]2[C@@H]2CC[C@H]([C@@H](CCC(O)=O)C)[C@@]2(C)[C@@H](O)C1 KXGVEGMKQFWNSR-LLQZFEROSA-N 0.000 description 1
- 229960003964 deoxycholic acid Drugs 0.000 description 1
- 230000001419 dependent effect Effects 0.000 description 1
- 239000008121 dextrose Substances 0.000 description 1
- NEFBYIFKOOEVPA-UHFFFAOYSA-K dicalcium phosphate Chemical compound [Ca+2].[Ca+2].[O-]P([O-])([O-])=O NEFBYIFKOOEVPA-UHFFFAOYSA-K 0.000 description 1
- 229940038472 dicalcium phosphate Drugs 0.000 description 1
- 229910000390 dicalcium phosphate Inorganic materials 0.000 description 1
- 235000014113 dietary fatty acids Nutrition 0.000 description 1
- NVJBFARDFTXOTO-UHFFFAOYSA-N diethyl sulfite Chemical compound CCOS(=O)OCC NVJBFARDFTXOTO-UHFFFAOYSA-N 0.000 description 1
- 230000001079 digestive effect Effects 0.000 description 1
- 238000006471 dimerization reaction Methods 0.000 description 1
- 125000000118 dimethyl group Chemical group [H]C([H])([H])* 0.000 description 1
- 125000003438 dodecyl group Chemical group [H]C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])* 0.000 description 1
- 239000002552 dosage form Substances 0.000 description 1
- 230000037437 driver mutation Effects 0.000 description 1
- 238000001647 drug administration Methods 0.000 description 1
- 238000009510 drug design Methods 0.000 description 1
- 229940088679 drug related substance Drugs 0.000 description 1
- 238000001035 drying Methods 0.000 description 1
- 239000008393 encapsulating agent Substances 0.000 description 1
- 239000002702 enteric coating Substances 0.000 description 1
- 238000009505 enteric coating Methods 0.000 description 1
- 230000002255 enzymatic effect Effects 0.000 description 1
- 238000001952 enzyme assay Methods 0.000 description 1
- 229960001433 erlotinib Drugs 0.000 description 1
- AAKJLRGGTJKAMG-UHFFFAOYSA-N erlotinib Chemical compound C=12C=C(OCCOC)C(OCCOC)=CC2=NC=NC=1NC1=CC=CC(C#C)=C1 AAKJLRGGTJKAMG-UHFFFAOYSA-N 0.000 description 1
- AFAXGSQYZLGZPG-UHFFFAOYSA-N ethanedisulfonic acid Chemical compound OS(=O)(=O)CCS(O)(=O)=O AFAXGSQYZLGZPG-UHFFFAOYSA-N 0.000 description 1
- CCIVGXIOQKPBKL-UHFFFAOYSA-M ethanesulfonate Chemical compound CCS([O-])(=O)=O CCIVGXIOQKPBKL-UHFFFAOYSA-M 0.000 description 1
- BEFDCLMNVWHSGT-UHFFFAOYSA-N ethenylcyclopentane Chemical compound C=CC1CCCC1 BEFDCLMNVWHSGT-UHFFFAOYSA-N 0.000 description 1
- ZMMJGEGLRURXTF-UHFFFAOYSA-N ethidium bromide Chemical compound [Br-].C12=CC(N)=CC=C2C2=CC=C(N)C=C2[N+](CC)=C1C1=CC=CC=C1 ZMMJGEGLRURXTF-UHFFFAOYSA-N 0.000 description 1
- 229960005542 ethidium bromide Drugs 0.000 description 1
- 229940093499 ethyl acetate Drugs 0.000 description 1
- LVGKNOAMLMIIKO-QXMHVHEDSA-N ethyl oleate Chemical compound CCCCCCCC\C=C/CCCCCCCC(=O)OCC LVGKNOAMLMIIKO-QXMHVHEDSA-N 0.000 description 1
- 229940093471 ethyl oleate Drugs 0.000 description 1
- 238000011156 evaluation Methods 0.000 description 1
- 230000001747 exhibiting effect Effects 0.000 description 1
- 238000000605 extraction Methods 0.000 description 1
- 239000003885 eye ointment Substances 0.000 description 1
- 229930195729 fatty acid Natural products 0.000 description 1
- 239000000194 fatty acid Substances 0.000 description 1
- 239000000945 filler Substances 0.000 description 1
- 239000012065 filter cake Substances 0.000 description 1
- 238000000684 flow cytometry Methods 0.000 description 1
- 238000001943 fluorescence-activated cell sorting Methods 0.000 description 1
- 229910052731 fluorine Inorganic materials 0.000 description 1
- 239000011737 fluorine Substances 0.000 description 1
- 125000001153 fluoro group Chemical group F* 0.000 description 1
- 235000019253 formic acid Nutrition 0.000 description 1
- 239000012737 fresh medium Substances 0.000 description 1
- 239000001530 fumaric acid Substances 0.000 description 1
- 229940125777 fusion inhibitor Drugs 0.000 description 1
- 239000007789 gas Substances 0.000 description 1
- 210000001035 gastrointestinal tract Anatomy 0.000 description 1
- 229960002584 gefitinib Drugs 0.000 description 1
- XGALLCVXEZPNRQ-UHFFFAOYSA-N gefitinib Chemical compound C=12C=C(OCCCN3CCOCC3)C(OC)=CC2=NC=NC=1NC1=CC=C(F)C(Cl)=C1 XGALLCVXEZPNRQ-UHFFFAOYSA-N 0.000 description 1
- 239000000499 gel Substances 0.000 description 1
- 230000009368 gene silencing by RNA Effects 0.000 description 1
- 208000016361 genetic disease Diseases 0.000 description 1
- 238000003205 genotyping method Methods 0.000 description 1
- 239000000174 gluconic acid Substances 0.000 description 1
- 235000012208 gluconic acid Nutrition 0.000 description 1
- 239000008103 glucose Substances 0.000 description 1
- 235000001727 glucose Nutrition 0.000 description 1
- ZDXPYRJPNDTMRX-UHFFFAOYSA-N glutamine Natural products OC(=O)C(N)CCC(N)=O ZDXPYRJPNDTMRX-UHFFFAOYSA-N 0.000 description 1
- ZEMPKEQAKRGZGQ-XOQCFJPHSA-N glycerol triricinoleate Natural products CCCCCC[C@@H](O)CC=CCCCCCCCC(=O)OC[C@@H](COC(=O)CCCCCCCC=CC[C@@H](O)CCCCCC)OC(=O)CCCCCCCC=CC[C@H](O)CCCCCC ZEMPKEQAKRGZGQ-XOQCFJPHSA-N 0.000 description 1
- 239000008187 granular material Substances 0.000 description 1
- 230000012010 growth Effects 0.000 description 1
- 229940093915 gynecological organic acid Drugs 0.000 description 1
- 229910052736 halogen Inorganic materials 0.000 description 1
- 150000002367 halogens Chemical class 0.000 description 1
- 238000010438 heat treatment Methods 0.000 description 1
- BXWNKGSJHAJOGX-UHFFFAOYSA-N hexadecan-1-ol Chemical compound CCCCCCCCCCCCCCCCO BXWNKGSJHAJOGX-UHFFFAOYSA-N 0.000 description 1
- HNDVDQJCIGZPNO-UHFFFAOYSA-N histidine Natural products OC(=O)C(N)CC1=CN=CN1 HNDVDQJCIGZPNO-UHFFFAOYSA-N 0.000 description 1
- 238000002657 hormone replacement therapy Methods 0.000 description 1
- 238000001794 hormone therapy Methods 0.000 description 1
- 102000054387 human GOPC Human genes 0.000 description 1
- 102000049336 human SLC34A2 Human genes 0.000 description 1
- 239000003906 humectant Substances 0.000 description 1
- 210000004408 hybridoma Anatomy 0.000 description 1
- 150000004677 hydrates Chemical class 0.000 description 1
- 230000007062 hydrolysis Effects 0.000 description 1
- 238000006460 hydrolysis reaction Methods 0.000 description 1
- 230000002209 hydrophobic effect Effects 0.000 description 1
- 125000002887 hydroxy group Chemical group [H]O* 0.000 description 1
- 229920003063 hydroxymethyl cellulose Polymers 0.000 description 1
- 229940031574 hydroxymethyl cellulose Drugs 0.000 description 1
- 210000000987 immune system Anatomy 0.000 description 1
- 238000003317 immunochromatography Methods 0.000 description 1
- 102000018358 immunoglobulin Human genes 0.000 description 1
- 238000003364 immunohistochemistry Methods 0.000 description 1
- 238000001114 immunoprecipitation Methods 0.000 description 1
- 238000001727 in vivo Methods 0.000 description 1
- 238000011065 in-situ storage Methods 0.000 description 1
- 238000010348 incorporation Methods 0.000 description 1
- 238000011534 incubation Methods 0.000 description 1
- 239000011261 inert gas Substances 0.000 description 1
- ZPNFWUPYTFPOJU-LPYSRVMUSA-N iniprol Chemical compound C([C@H]1C(=O)NCC(=O)NCC(=O)N[C@H]2CSSC[C@H]3C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](C)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@H](C(N[C@H](C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC=4C=CC(O)=CC=4)C(=O)N[C@@H](CC=4C=CC=CC=4)C(=O)N[C@@H](CC=4C=CC(O)=CC=4)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](C)C(=O)NCC(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CSSC[C@H](NC(=O)[C@H](CC(O)=O)NC(=O)[C@H](CCC(O)=O)NC(=O)[C@H](C)NC(=O)[C@H](CO)NC(=O)[C@H](CCCCN)NC(=O)[C@H](CC=4C=CC=CC=4)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@H](CCCNC(N)=N)NC(=O)[C@H](CCCCN)NC(=O)[C@H](C)NC(=O)[C@H](CCCNC(N)=N)NC2=O)C(=O)N[C@@H](CCSC)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CSSC[C@H](NC(=O)[C@H](CC=2C=CC=CC=2)NC(=O)[C@H](CC(O)=O)NC(=O)[C@H]2N(CCC2)C(=O)[C@@H](N)CCCNC(N)=N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCC(O)=O)C(=O)N2[C@@H](CCC2)C(=O)N2[C@@H](CCC2)C(=O)N[C@@H](CC=2C=CC(O)=CC=2)C(=O)N[C@@H]([C@@H](C)O)C(=O)NCC(=O)N2[C@@H](CCC2)C(=O)N3)C(=O)NCC(=O)NCC(=O)N[C@@H](C)C(O)=O)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@H](C(=O)N[C@@H](CC=2C=CC=CC=2)C(=O)N[C@H](C(=O)N1)C(C)C)[C@@H](C)O)[C@@H](C)CC)=O)[C@@H](C)CC)C1=CC=C(O)C=C1 ZPNFWUPYTFPOJU-LPYSRVMUSA-N 0.000 description 1
- 238000002347 injection Methods 0.000 description 1
- 239000007924 injection Substances 0.000 description 1
- 238000003780 insertion Methods 0.000 description 1
- 230000037431 insertion Effects 0.000 description 1
- 230000010354 integration Effects 0.000 description 1
- 230000003993 interaction Effects 0.000 description 1
- 238000007918 intramuscular administration Methods 0.000 description 1
- 238000001990 intravenous administration Methods 0.000 description 1
- 238000011835 investigation Methods 0.000 description 1
- 239000011630 iodine Chemical group 0.000 description 1
- 229940036571 iodine therapy Drugs 0.000 description 1
- 238000002955 isolation Methods 0.000 description 1
- 229960000310 isoleucine Drugs 0.000 description 1
- AGPKZVBTJJNPAG-UHFFFAOYSA-N isoleucine Natural products CCC(C)C(N)C(O)=O AGPKZVBTJJNPAG-UHFFFAOYSA-N 0.000 description 1
- 150000002540 isothiocyanates Chemical class 0.000 description 1
- 239000007951 isotonicity adjuster Substances 0.000 description 1
- NLYAJNPCOHFWQQ-UHFFFAOYSA-N kaolin Chemical compound O.O.O=[Al]O[Si](=O)O[Si](=O)O[Al]=O NLYAJNPCOHFWQQ-UHFFFAOYSA-N 0.000 description 1
- 239000004310 lactic acid Substances 0.000 description 1
- 235000014655 lactic acid Nutrition 0.000 description 1
- 239000008101 lactose Substances 0.000 description 1
- 235000010445 lecithin Nutrition 0.000 description 1
- 239000000787 lecithin Substances 0.000 description 1
- 229940067606 lecithin Drugs 0.000 description 1
- 210000004901 leucine-rich repeat Anatomy 0.000 description 1
- GDBQQVLCIARPGH-ULQDDVLXSA-N leupeptin Chemical compound CC(C)C[C@H](NC(C)=O)C(=O)N[C@@H](CC(C)C)C(=O)N[C@H](C=O)CCCN=C(N)N GDBQQVLCIARPGH-ULQDDVLXSA-N 0.000 description 1
- 108010052968 leupeptin Proteins 0.000 description 1
- 230000000670 limiting effect Effects 0.000 description 1
- 201000007270 liver cancer Diseases 0.000 description 1
- 239000000314 lubricant Substances 0.000 description 1
- 238000004020 luminiscence type Methods 0.000 description 1
- 201000009546 lung large cell carcinoma Diseases 0.000 description 1
- 201000005243 lung squamous cell carcinoma Diseases 0.000 description 1
- 230000001926 lymphatic effect Effects 0.000 description 1
- 239000008176 lyophilized powder Substances 0.000 description 1
- 239000012139 lysis buffer Substances 0.000 description 1
- 238000007403 mPCR Methods 0.000 description 1
- 229920002521 macromolecule Polymers 0.000 description 1
- VZCYOOQTPOCHFL-UPHRSURJSA-N maleic acid Chemical compound OC(=O)\C=C/C(O)=O VZCYOOQTPOCHFL-UPHRSURJSA-N 0.000 description 1
- 239000011976 maleic acid Substances 0.000 description 1
- 239000001630 malic acid Substances 0.000 description 1
- 229940099690 malic acid Drugs 0.000 description 1
- 229960002510 mandelic acid Drugs 0.000 description 1
- 239000000594 mannitol Substances 0.000 description 1
- 235000010355 mannitol Nutrition 0.000 description 1
- 239000003550 marker Substances 0.000 description 1
- 238000004949 mass spectrometry Methods 0.000 description 1
- 229940127554 medical product Drugs 0.000 description 1
- 201000001441 melanoma Diseases 0.000 description 1
- 229910052751 metal Inorganic materials 0.000 description 1
- 239000002184 metal Substances 0.000 description 1
- 229940098779 methanesulfonic acid Drugs 0.000 description 1
- 229930182817 methionine Natural products 0.000 description 1
- 229920000609 methyl cellulose Polymers 0.000 description 1
- 125000002496 methyl group Chemical group [H]C([H])([H])* 0.000 description 1
- WBYWAXJHAXSJNI-UHFFFAOYSA-N methyl p-hydroxycinnamate Natural products OC(=O)C=CC1=CC=CC=C1 WBYWAXJHAXSJNI-UHFFFAOYSA-N 0.000 description 1
- 239000001923 methylcellulose Substances 0.000 description 1
- GRVDJDISBSALJP-UHFFFAOYSA-N methyloxidanyl Chemical group [O]C GRVDJDISBSALJP-UHFFFAOYSA-N 0.000 description 1
- 108091070501 miRNA Proteins 0.000 description 1
- 239000002679 microRNA Substances 0.000 description 1
- 238000002493 microarray Methods 0.000 description 1
- 244000005700 microbiome Species 0.000 description 1
- 229940016286 microcrystalline cellulose Drugs 0.000 description 1
- 235000019813 microcrystalline cellulose Nutrition 0.000 description 1
- 239000008108 microcrystalline cellulose Substances 0.000 description 1
- 108010029942 microperoxidase Proteins 0.000 description 1
- 150000007522 mineralic acids Chemical class 0.000 description 1
- 239000003607 modifier Substances 0.000 description 1
- CQDGTJPVBWZJAZ-UHFFFAOYSA-N monoethyl carbonate Chemical compound CCOC(O)=O CQDGTJPVBWZJAZ-UHFFFAOYSA-N 0.000 description 1
- 229940126619 mouse monoclonal antibody Drugs 0.000 description 1
- 239000002105 nanoparticle Substances 0.000 description 1
- KVBGVZZKJNLNJU-UHFFFAOYSA-N naphthalene-2-sulfonic acid Chemical compound C1=CC=CC2=CC(S(=O)(=O)O)=CC=C21 KVBGVZZKJNLNJU-UHFFFAOYSA-N 0.000 description 1
- 239000013642 negative control Substances 0.000 description 1
- 229910017604 nitric acid Inorganic materials 0.000 description 1
- 231100000344 non-irritating Toxicity 0.000 description 1
- 239000012457 nonaqueous media Substances 0.000 description 1
- 239000002687 nonaqueous vehicle Substances 0.000 description 1
- 231100000252 nontoxic Toxicity 0.000 description 1
- 230000003000 nontoxic effect Effects 0.000 description 1
- 238000007899 nucleic acid hybridization Methods 0.000 description 1
- QIQXTHQIDYTFRH-UHFFFAOYSA-N octadecanoic acid Chemical compound CCCCCCCCCCCCCCCCCC(O)=O QIQXTHQIDYTFRH-UHFFFAOYSA-N 0.000 description 1
- OQCDKBAXFALNLD-UHFFFAOYSA-N octadecanoic acid Natural products CCCCCCCC(C)CCCCCCCCC(O)=O OQCDKBAXFALNLD-UHFFFAOYSA-N 0.000 description 1
- 239000002674 ointment Substances 0.000 description 1
- 230000004650 oncogenic pathway Effects 0.000 description 1
- 238000011275 oncology therapy Methods 0.000 description 1
- 210000000056 organ Anatomy 0.000 description 1
- 150000007524 organic acids Chemical class 0.000 description 1
- 235000005985 organic acids Nutrition 0.000 description 1
- 150000002895 organic esters Chemical class 0.000 description 1
- 229940026778 other chemotherapeutics in atc Drugs 0.000 description 1
- 235000006408 oxalic acid Nutrition 0.000 description 1
- 239000006179 pH buffering agent Substances 0.000 description 1
- 208000021010 pancreatic neuroendocrine tumor Diseases 0.000 description 1
- 239000012188 paraffin wax Substances 0.000 description 1
- 230000037361 pathway Effects 0.000 description 1
- 239000013610 patient sample Substances 0.000 description 1
- 239000000312 peanut oil Substances 0.000 description 1
- 108010091212 pepstatin Proteins 0.000 description 1
- FAXGPCHRFPCXOO-LXTPJMTPSA-N pepstatin A Chemical compound OC(=O)C[C@H](O)[C@H](CC(C)C)NC(=O)[C@H](C)NC(=O)C[C@H](O)[C@H](CC(C)C)NC(=O)[C@H](C(C)C)NC(=O)[C@H](C(C)C)NC(=O)CC(C)C FAXGPCHRFPCXOO-LXTPJMTPSA-N 0.000 description 1
- 238000002823 phage display Methods 0.000 description 1
- 229960003742 phenol Drugs 0.000 description 1
- WVDDGKGOMKODPV-ZQBYOMGUSA-N phenyl(114C)methanol Chemical compound O[14CH2]C1=CC=CC=C1 WVDDGKGOMKODPV-ZQBYOMGUSA-N 0.000 description 1
- 235000021317 phosphate Nutrition 0.000 description 1
- 150000003013 phosphoric acid derivatives Chemical class 0.000 description 1
- 239000002504 physiological saline solution Substances 0.000 description 1
- IUGYQRQAERSCNH-UHFFFAOYSA-N pivalic acid Chemical compound CC(C)(C)C(O)=O IUGYQRQAERSCNH-UHFFFAOYSA-N 0.000 description 1
- 229910052697 platinum Inorganic materials 0.000 description 1
- 229920002647 polyamide Polymers 0.000 description 1
- 229920000642 polymer Polymers 0.000 description 1
- 229920005862 polyol Polymers 0.000 description 1
- 150000003077 polyols Chemical class 0.000 description 1
- 239000001267 polyvinylpyrrolidone Substances 0.000 description 1
- 235000013855 polyvinylpyrrolidone Nutrition 0.000 description 1
- 229920000036 polyvinylpyrrolidone Polymers 0.000 description 1
- 239000013641 positive control Substances 0.000 description 1
- WFIZEGIEIOHZCP-UHFFFAOYSA-M potassium formate Chemical compound [K+].[O-]C=O WFIZEGIEIOHZCP-UHFFFAOYSA-M 0.000 description 1
- WSHYKIAQCMIPTB-UHFFFAOYSA-M potassium;2-oxo-3-(3-oxo-1-phenylbutyl)chromen-4-olate Chemical compound [K+].[O-]C=1C2=CC=CC=C2OC(=O)C=1C(CC(=O)C)C1=CC=CC=C1 WSHYKIAQCMIPTB-UHFFFAOYSA-M 0.000 description 1
- 239000002244 precipitate Substances 0.000 description 1
- 150000003140 primary amides Chemical class 0.000 description 1
- VYXXMAGSIYIYGD-NWAYQTQBSA-N propan-2-yl 2-[[[(2R)-1-(6-aminopurin-9-yl)propan-2-yl]oxymethyl-(pyrimidine-4-carbonylamino)phosphoryl]amino]-2-methylpropanoate Chemical compound CC(C)OC(=O)C(C)(C)NP(=O)(CO[C@H](C)Cn1cnc2c(N)ncnc12)NC(=O)c1ccncn1 VYXXMAGSIYIYGD-NWAYQTQBSA-N 0.000 description 1
- 239000003380 propellant Substances 0.000 description 1
- 235000019260 propionic acid Nutrition 0.000 description 1
- 108020001580 protein domains Proteins 0.000 description 1
- 229940107700 pyruvic acid Drugs 0.000 description 1
- 150000003856 quaternary ammonium compounds Chemical class 0.000 description 1
- IUVKMZGDUIUOCP-BTNSXGMBSA-N quinbolone Chemical compound O([C@H]1CC[C@H]2[C@H]3[C@@H]([C@]4(C=CC(=O)C=C4CC3)C)CC[C@@]21C)C1=CCCC1 IUVKMZGDUIUOCP-BTNSXGMBSA-N 0.000 description 1
- 125000004549 quinolin-4-yl group Chemical group N1=CC=C(C2=CC=CC=C12)* 0.000 description 1
- 230000005855 radiation Effects 0.000 description 1
- 230000002285 radioactive effect Effects 0.000 description 1
- 239000000941 radioactive substance Substances 0.000 description 1
- 238000001959 radiotherapy Methods 0.000 description 1
- 229940124617 receptor tyrosine kinase inhibitor Drugs 0.000 description 1
- 238000001953 recrystallisation Methods 0.000 description 1
- 230000002829 reductive effect Effects 0.000 description 1
- 238000010992 reflux Methods 0.000 description 1
- 238000007894 restriction fragment length polymorphism technique Methods 0.000 description 1
- 230000000717 retained effect Effects 0.000 description 1
- 238000010839 reverse transcription Methods 0.000 description 1
- 239000012723 sample buffer Substances 0.000 description 1
- 238000003118 sandwich ELISA Methods 0.000 description 1
- 229920006395 saturated elastomer Polymers 0.000 description 1
- 230000007017 scission Effects 0.000 description 1
- 238000012106 screening analysis Methods 0.000 description 1
- 238000007423 screening assay Methods 0.000 description 1
- 238000000926 separation method Methods 0.000 description 1
- 239000008159 sesame oil Substances 0.000 description 1
- 235000011803 sesame oil Nutrition 0.000 description 1
- 230000019491 signal transduction Effects 0.000 description 1
- 230000008054 signal transmission Effects 0.000 description 1
- 230000011664 signaling Effects 0.000 description 1
- 239000002924 silencing RNA Substances 0.000 description 1
- 150000004760 silicates Chemical class 0.000 description 1
- RMAQACBXLXPBSY-UHFFFAOYSA-N silicic acid Chemical compound O[Si](O)(O)O RMAQACBXLXPBSY-UHFFFAOYSA-N 0.000 description 1
- 239000000377 silicon dioxide Substances 0.000 description 1
- 235000012239 silicon dioxide Nutrition 0.000 description 1
- 238000004088 simulation Methods 0.000 description 1
- 239000002002 slurry Substances 0.000 description 1
- 239000004055 small Interfering RNA Substances 0.000 description 1
- 208000000649 small cell carcinoma Diseases 0.000 description 1
- 235000010413 sodium alginate Nutrition 0.000 description 1
- 239000000661 sodium alginate Substances 0.000 description 1
- 229940005550 sodium alginate Drugs 0.000 description 1
- 229910000029 sodium carbonate Inorganic materials 0.000 description 1
- 239000001509 sodium citrate Substances 0.000 description 1
- NLJMYIDDQXHKNR-UHFFFAOYSA-K sodium citrate Chemical compound O.O.[Na+].[Na+].[Na+].[O-]C(=O)CC(O)(CC([O-])=O)C([O-])=O NLJMYIDDQXHKNR-UHFFFAOYSA-K 0.000 description 1
- FQENQNTWSFEDLI-UHFFFAOYSA-J sodium diphosphate Chemical compound [Na+].[Na+].[Na+].[Na+].[O-]P([O-])(=O)OP([O-])([O-])=O FQENQNTWSFEDLI-UHFFFAOYSA-J 0.000 description 1
- 229940048086 sodium pyrophosphate Drugs 0.000 description 1
- CGRKYEALWSRNJS-UHFFFAOYSA-N sodium;2-methylbutan-2-olate Chemical compound [Na+].CCC(C)(C)[O-] CGRKYEALWSRNJS-UHFFFAOYSA-N 0.000 description 1
- HPALAKNZSZLMCH-UHFFFAOYSA-M sodium;chloride;hydrate Chemical compound O.[Na+].[Cl-] HPALAKNZSZLMCH-UHFFFAOYSA-M 0.000 description 1
- 239000007909 solid dosage form Substances 0.000 description 1
- 230000003381 solubilizing effect Effects 0.000 description 1
- 239000004334 sorbic acid Substances 0.000 description 1
- 235000010199 sorbic acid Nutrition 0.000 description 1
- 229940075582 sorbic acid Drugs 0.000 description 1
- 239000000600 sorbitol Substances 0.000 description 1
- 230000009870 specific binding Effects 0.000 description 1
- 238000011895 specific detection Methods 0.000 description 1
- 230000003595 spectral effect Effects 0.000 description 1
- 239000007921 spray Substances 0.000 description 1
- 208000024794 sputum Diseases 0.000 description 1
- 210000003802 sputum Anatomy 0.000 description 1
- 238000010186 staining Methods 0.000 description 1
- 239000008117 stearic acid Substances 0.000 description 1
- 238000003756 stirring Methods 0.000 description 1
- 238000007920 subcutaneous administration Methods 0.000 description 1
- 235000000346 sugar Nutrition 0.000 description 1
- 150000008163 sugars Chemical class 0.000 description 1
- 230000001629 suppression Effects 0.000 description 1
- 238000001356 surgical procedure Methods 0.000 description 1
- 230000004083 survival effect Effects 0.000 description 1
- 238000003786 synthesis reaction Methods 0.000 description 1
- 239000006188 syrup Substances 0.000 description 1
- 235000020357 syrup Nutrition 0.000 description 1
- 239000000454 talc Substances 0.000 description 1
- 229910052623 talc Inorganic materials 0.000 description 1
- 235000012222 talc Nutrition 0.000 description 1
- 238000002626 targeted therapy Methods 0.000 description 1
- 230000008685 targeting Effects 0.000 description 1
- 239000011975 tartaric acid Substances 0.000 description 1
- 235000002906 tartaric acid Nutrition 0.000 description 1
- BSYVTEYKTMYBMK-UHFFFAOYSA-N tetrahydrofurfuryl alcohol Chemical compound OCC1CCCO1 BSYVTEYKTMYBMK-UHFFFAOYSA-N 0.000 description 1
- 235000019818 tetrasodium diphosphate Nutrition 0.000 description 1
- 239000001577 tetrasodium phosphonato phosphate Substances 0.000 description 1
- 150000003573 thiols Chemical class 0.000 description 1
- 201000002510 thyroid cancer Diseases 0.000 description 1
- 238000011200 topical administration Methods 0.000 description 1
- 230000000699 topical effect Effects 0.000 description 1
- 230000002103 transcriptional effect Effects 0.000 description 1
- 238000012546 transfer Methods 0.000 description 1
- 230000001131 transforming effect Effects 0.000 description 1
- 229960000575 trastuzumab Drugs 0.000 description 1
- 229940117013 triethanolamine oleate Drugs 0.000 description 1
- IHIXIJGXTJIKRB-UHFFFAOYSA-N trisodium vanadate Chemical compound [Na+].[Na+].[Na+].[O-][V]([O-])([O-])=O IHIXIJGXTJIKRB-UHFFFAOYSA-N 0.000 description 1
- 125000000430 tryptophan group Chemical group [H]N([H])C(C(=O)O*)C([H])([H])C1=C([H])N([H])C2=C([H])C([H])=C([H])C([H])=C12 0.000 description 1
- 230000004614 tumor growth Effects 0.000 description 1
- OUYCCCASQSFEME-UHFFFAOYSA-N tyrosine Natural products OC(=O)C(N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-UHFFFAOYSA-N 0.000 description 1
- 210000002700 urine Anatomy 0.000 description 1
- 239000004474 valine Substances 0.000 description 1
- 210000005166 vasculature Anatomy 0.000 description 1
- 235000015112 vegetable and seed oil Nutrition 0.000 description 1
- 239000008158 vegetable oil Substances 0.000 description 1
- 239000011534 wash buffer Substances 0.000 description 1
- 238000009736 wetting Methods 0.000 description 1
Classifications
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K31/00—Medicinal preparations containing organic active ingredients
- A61K31/33—Heterocyclic compounds
- A61K31/395—Heterocyclic compounds having nitrogen as a ring hetero atom, e.g. guanethidine or rifamycins
- A61K31/435—Heterocyclic compounds having nitrogen as a ring hetero atom, e.g. guanethidine or rifamycins having six-membered rings with one nitrogen as the only ring hetero atom
- A61K31/47—Quinolines; Isoquinolines
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K31/00—Medicinal preparations containing organic active ingredients
- A61K31/33—Heterocyclic compounds
- A61K31/335—Heterocyclic compounds having oxygen as the only ring hetero atom, e.g. fungichromin
- A61K31/337—Heterocyclic compounds having oxygen as the only ring hetero atom, e.g. fungichromin having four-membered rings, e.g. taxol
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K31/00—Medicinal preparations containing organic active ingredients
- A61K31/33—Heterocyclic compounds
- A61K31/395—Heterocyclic compounds having nitrogen as a ring hetero atom, e.g. guanethidine or rifamycins
- A61K31/495—Heterocyclic compounds having nitrogen as a ring hetero atom, e.g. guanethidine or rifamycins having six-membered rings with two or more nitrogen atoms as the only ring heteroatoms, e.g. piperazine or tetrazines
- A61K31/505—Pyrimidines; Hydrogenated pyrimidines, e.g. trimethoprim
- A61K31/517—Pyrimidines; Hydrogenated pyrimidines, e.g. trimethoprim ortho- or peri-condensed with carbocyclic ring systems, e.g. quinazoline, perimidine
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K31/00—Medicinal preparations containing organic active ingredients
- A61K31/70—Carbohydrates; Sugars; Derivatives thereof
- A61K31/7042—Compounds having saccharide radicals and heterocyclic rings
- A61K31/7052—Compounds having saccharide radicals and heterocyclic rings having nitrogen as a ring hetero atom, e.g. nucleosides, nucleotides
- A61K31/706—Compounds having saccharide radicals and heterocyclic rings having nitrogen as a ring hetero atom, e.g. nucleosides, nucleotides containing six-membered rings with nitrogen as a ring hetero atom
- A61K31/7064—Compounds having saccharide radicals and heterocyclic rings having nitrogen as a ring hetero atom, e.g. nucleosides, nucleotides containing six-membered rings with nitrogen as a ring hetero atom containing condensed or non-condensed pyrimidines
- A61K31/7068—Compounds having saccharide radicals and heterocyclic rings having nitrogen as a ring hetero atom, e.g. nucleosides, nucleotides containing six-membered rings with nitrogen as a ring hetero atom containing condensed or non-condensed pyrimidines having oxo groups directly attached to the pyrimidine ring, e.g. cytidine, cytidylic acid
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P11/00—Drugs for disorders of the respiratory system
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P43/00—Drugs for specific purposes, not provided for in groups A61P1/00-A61P41/00
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6876—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes
- C12Q1/6883—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes for diseases caused by alterations of genetic material
- C12Q1/6886—Nucleic acid products used in the analysis of nucleic acids, e.g. primers or probes for diseases caused by alterations of genetic material for cancer
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K2300/00—Mixtures or combinations of active ingredients, wherein at least one active ingredient is fully defined in groups A61K31/00 - A61K41/00
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/158—Expression markers
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Chemical & Material Sciences (AREA)
- General Health & Medical Sciences (AREA)
- Medicinal Chemistry (AREA)
- Pharmacology & Pharmacy (AREA)
- Animal Behavior & Ethology (AREA)
- Public Health (AREA)
- Veterinary Medicine (AREA)
- Epidemiology (AREA)
- Organic Chemistry (AREA)
- Engineering & Computer Science (AREA)
- Molecular Biology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Nuclear Medicine, Radiotherapy & Molecular Imaging (AREA)
- Chemical Kinetics & Catalysis (AREA)
- General Chemical & Material Sciences (AREA)
- Immunology (AREA)
- Wood Science & Technology (AREA)
- Pathology (AREA)
- Genetics & Genomics (AREA)
- Analytical Chemistry (AREA)
- Zoology (AREA)
- Microbiology (AREA)
- Biophysics (AREA)
- Biotechnology (AREA)
- Oncology (AREA)
- Physics & Mathematics (AREA)
- Hospice & Palliative Care (AREA)
- Biochemistry (AREA)
- General Engineering & Computer Science (AREA)
- Pulmonology (AREA)
- Pharmaceuticals Containing Other Organic And Inorganic Compounds (AREA)
- Acyclic And Carbocyclic Compounds In Medicinal Compositions (AREA)
- Measuring Or Testing Involving Enzymes Or Micro-Organisms (AREA)
- Medicines That Contain Protein Lipid Enzymes And Other Medicines (AREA)
Abstract
Compuesto para su uso en un procedimiento de tratamiento de adenocarcinoma de pulmón en un paciente, en el que el compuesto es el compuesto 1: **(Ver fórmula)** o una sal farmacéuticamente aceptable del mismo; y en el que el adenocarcinoma de pulmón es cáncer de pulmón de células no pequeñas positivo en la fusión de SLC34A2-ROS1, CD74-ROS1 o FIG-ROS1.
Description
DESCRIPCIÓN
Procedimiento de tratamiento de adenocarcinoma de pulmón
Listado de secuencia
[0001] El listado de secuencia titulado "EX14-003C-PC_2015_04_20_SEQUENCE_LISTING_ST25.txt," creado el 27 de abril de 2015, a las 12:54, que tiene 26 KB, está presentado electrónicamente con el presente documento.
Campo de la Invención
[0002] La presente invención está dirigida a un inhibidor de receptor de tirosina quinasas para el uso en la detección, el diagnóstico y el tratamiento de cáncer, en particular, del adenocarcinoma de pulmón.
Antecedentes de la Invención
[0003] El cáncer de pulmón es la causa principal de mortalidad relacionada con el cáncer a nivel mundial. Los desarrollos recientes en terapias dirigidas han llevado a un cambio de paradigma en el tratamiento de cáncer de pulmón de células no pequeñas (NSCLC). Véanse, Mok TS, Wu YL, Thongprasert S, Yang CH, Chu DT, Saijo N, Sunpaweravong P, Han B, Margono B, Ichinose Y, Nishiwaki Y, Ohe Y, Yang JJ, Chewaskulyong B, Jiang H, Duffield EL, Watkins CL, Armour AA, Fukuoka M: "Gefitinib or carboplatin-paclitaxel in pulmonary adenocarcinoma", N Engl J Med. 3 de Septiembre de 2009;361(10):947-57; Maemondo M, Inoue A, Kobayashi K, Sugawara S, Oizumi S, Isobe H, Gemma A, Harada M, Yoshizawa H, Kinoshita I, Fujita Y, Okinaga S, Hirano H, Yoshimori K, Harada T, Ogura T, Ando M, Miyazawa H, Tanaka T, Saijo Y, Hagiwara K, Morita S, Nukiwa T; North-East Japan Study Group: "Gefitinib or chemotherapy for non-small-cell lung cancer with mutated EGFR", N Engl J Med. 24 de junio de 2010; 362(25): 2380-8. Los inhibidores de tirosina quinasa (TKI) del receptor de factor de crecimiento epidérmico (EGFR), gefitinib y erlotinib, y los TKI de la quinasa del linfoma anaplásico (ALK), crizotinib, han mostrado actividad clínica en pacientes de NSCLC con mutaciones de EGFR o reorganizaciones genéticas de ALK. Véanse, Kwak EL, Bang YJ, Camidge DR, Shaw AT, Solomon B, Maki RG, Ou SH, Dezube BJ, Janne PA, Costa DB, Varella-Garcia M, Kim WH, Lynch TJ, Fidias P, Stubbs H, Engelman JA, Sequist LV, Tan W, Gandhi L, Mino-Kenudson M, Wei GC, Shreeve SM, Ratain MJ, Settleman J, Christensen JG, Haber Da , Wilner K, Salgia R, Shapiro GI, Clark JW, Iafrate AJ: "Anaplastic lymphoma kinase inhibition in non-small-cell lung cancer", N Engl J Med. 28 de Octubre de 2010; 363(18):1693-703; Shaw AT, Camidge, Engelman JA, Solomon BJ, Kwak EL, Clark JW, Salgia R, Shapiro, Bang YJ, Tan W, Tye L, Wilner KD, Stephenson P, Varella-Garcia M, Bergethon K, Iafrate AJ, Ou SH: "Clinical activity of crizotinib in advanced non-small cell lung cancer (NSCLC) harboring ROS1 gene rearrangement", J Clin Oncol. 2012 30 (suppl; abstr 7508). La fusión del gen KIF5B (la familia de quinesina 5B) y el oncogén RET se han descrito recientemente como una mutación impulsora en 1-2% de pacientes de NSCLC y son un foco como una diana terapéutica. Véanse, Kohno T, Ichikawa H, Totoki Y, Yasuda K, Hiramoto M, Nammo T, Sakamoto H, Tsuta K, Furuta K, Shimada Y, Iwakawa 35 R, Ogiwara H, Oike T, Enari M, Schetter AJ, Okayama H, Haugen A, Skaug V, Chiku S, Yamanaka I, Arai Y, Watanabe S, Sekine I, Ogawa S, Harris CC, Tsuda H, Yoshida T, Yokota J, Shibata T: "KIF5B-RET fusions in lung adenocarcinoma", Nat Med. 12 de febrero de 2012; 18(3): 375-7; Takeuchi K, Soda M, Togashi Y, Suzuki R, Sakata S, Hatano S, Asaka R, Hamanaka W, Ninomiya H, Uehara H, Lim Choi Y, Satoh Y, Okumura S, Nakagawa K, Mano H, Ishikawa Y: "RET, ROS1 and ALK fusions in lung cancer", Nat Med. 12 de Febrero de 2012; 18(3): 378-81; Lipson D, Capelletti M, Yelensky R, Otto G, Parker A, Jarosz M, Curran JA, Balasubramanian S, Bloom T, Brennan KW, Donahue A, Downing SR, Frampton GM, Garcia L, Juhn F, Mitchell KC, White E, White J, Zwirko Z, Peretz T, Nechushtan H, Soussan-Gutman L, Kim J, Sasaki H, Kim HR, Park SI, Ercan D, Sheehan CE, Ross JS, Cronin MT, Janne PA, Stephens PJ: "Identification of new ALK and RET gene fusions from colorectal and lung cancer biopsies", Nat Med. 12 de febrero de 2012; 18(3): 382-4. Por lo tanto, cada vez es más importante identificar los genes impulsores clave en NSCLC y desarrollar terapias para cada subgrupo genómico de pacientes.
[0004] El documento WO 2014/039971 A1 da a conocer el tratamiento de adenocarcinoma de pulmón con un inhibidor de MET, VEGF y RET, según la Formula I como se muestra en el presente documento a continuación.
[0005] Kohno T, Tsuta K, Tsuchihara K, Nakoku T, Yoh K, Goto K, "RET fusion gene: Translation to personalized lung cancer therapy", Cancer Sci Noviembre de 2013; 104(11): 1396-1400 divulga que pruebas experimentales clínicas están en progreso para investigar los efectos terapéuticos de inhibidores de tirosina quinasa RET en pacientes con cáncer de pulmón de células no pequeñas positivo en la fusión de RET.
[0006] Drilon A, Wang L, Hasanovic A, Suehara Y, Lipson D, Stephens PJ, Ross J, Miller VA, Ginsberg MS, Zakowski MF, Kris MG, Ladanyi M, Rizvi NA "Response to Cabozantinib in Patients with RET Fusion-Positive Lung Adenocarcinomas", Cancer Discov., Junio, 630-635 presenta datos preliminares para los primeros tres pacientes tratados con el inhibidor RET de cabozantinib en una posible fase II de prueba experimental para pacientes con NSCLC positivo en la fusión de RET.
Características de la Invención
[0007] Estas y otras necesidades se cumplen en la presente invención que está dirigida a un compuesto para el uso en un procedimiento de tratamiento de adenocarcinoma de pulmón en un paciente, donde el compuesto es un compuesto 1:
o una sal farmacéuticamente aceptable del mismo; y donde el adenocarcinoma de pulmón es cáncer de pulmón de células no pequeñas positivo en la fusión de SLC34A2-ROS1, CD74-ROS1 o FIG-ROS1. La presente invención está dirigida también a un compuesto para el uso en un procedimiento de diagnóstico y tratamiento de un paciente, donde el paciente tiene un tumor NSCLC y el tumor se identifica como NSCLC positivo en la fusión de SLC34A2-ROS1, CD74-ROS1 o FIG-ROS1, donde el compuesto es el compuesto 1 o una sal farmacéuticamente aceptable del mismo y donde el compuesto se administra con al menos un portador farmacéuticamente aceptable.
[0008] Más generalmente, en el presente documento se da a conocer un procedimiento para tratar adenocarcinoma de pulmón utilizando un inhibidor de actividad de tirosina quinasa, especialmente, actividad de quinasa ROS1. El procedimiento comprende administrar una cantidad terapéuticamente eficaz de un compuesto o una sal farmacéuticamente aceptable del mismo que modula la quinasa ROS1 y/o una proteína de fusión de ROS1 en un paciente que necesita tal tratamiento. En una realización, el adenocarcinoma de pulmón es cáncer de pulmón de células no pequeñas (NSCLC). Más particularmente, el adenocarcinoma de pulmón es más frecuentemente NSCLC positivo en la fusión de SLC34a 2-r Os 1, NSCLC positivo en la fusión de c D74-ROS1, NSCLC positivo en la fusión de FIG-ROS1 (por ejemplo, CD74-ROS1, FIG-ROS1, FIG-ROS1(L), FIG-ROS1(S) y FIG-ROS1(VL)) y NSCLC que alberga otras proteínas de fusión de ROS1 conocidas. La quinasa ROS1 es codificada por el gen de ROS1 en cromosoma 6 en humanos (6q22).
[0009] En un aspecto, la presente divulgación está dirigida a un procedimiento para tratar NSCLC en un paciente que necesita tal tratamiento, que comprende administrar una cantidad terapéuticamente eficaz de un compuesto o una sal farmacéuticamente aceptable del mismo que al mismo tiempo modula ROS1 y/o una proteína de fusión de ROS1 al el paciente.
[0010] En una realización de este y otros aspectos de la divulgación, el inhibidor de quinasa ROS1 o inhibidor de quinasa de fusión ROS1 es un compuesto de Fórmula (I):
o una sal farmacéuticamente aceptable del mismo, donde:
R1 es halo;
R2 es halo;
R3 es alquilo (C1-C6);
R4 es alquilo (C1-C6); y
Q es CH o N.
[0011] En otra realización de la divulgación, el compuesto de la Fórmula I es un compuesto de Fórmula la
o una sal farmacéuticamente aceptable del mismo, donde:
R1 es halo;
R2 es halo; y
Q es CH o N.
[0012] En otra realización de la divulgación, el compuesto de la Fórmula I es compuesto 1:
o una sal farmacéuticamente aceptable del mismo. El compuesto 1 se conoce como N-(4-{[6,7-bis(metiloxi)quinolin-4-il]oxi}fenil)-N'-(4-fluorofenil)ciclopropano-1,1-dicarboxamida y por el nombre Cabozantinib. El 29 de noviembre de 2012, la sal de S-malato (es decir, sal de L-malato) de N-{4-[(6,7-dimetoxiquinolin-4-il)oxi]fenil}-N'-(4-fluorofenil) ciclopropano-1,1-dicarboxamida (también conocida como cabozantinib o COMETRIQ®) se aprobó por la Administración de Alimentos y Medicamentos de Estados Unidos para el tratamiento de cáncer de tiroides metastásico medular progresivo (MTC). En diciembre de 2013, el Comité Europeo de Productos Médicos para Uso Humano (CHMP), publicó una opinión positiva sobre la Solicitud de Autorización de Comercialización (MAA), aprobada por la Agencia Europea del Medicamento, o EMA, para COMETRIQ para la indicación propuesta de MTC progresivo, no resecable, avanzado localmente o metastásico. El Cabozantinib se está evaluando en un amplio programa de desarrollo, que incluye pruebas experimentales esenciales en fase 3 de desarrollo en cáncer de células renales metastásicas (RCC) y cáncer hepatocelular avanzado (HCC).
[0013] El compuesto 1 es un potente inhibidor de c-MET, RET y VEGFR2. Véanse, Yakes FM, Chen J, Tan J, Yamaguchi K, Shi Y, Yu P, Qian F, Chu F, Bentzien F, Cancilla B, Orf J, You A, Laird AD, Engst S, Lee L, Lesch J, Chou YC, Joly AH: "Cabozantinib (XL184), a novel MET and VEGFR2 inhibitor, simultaneously suppresses metastasis, angiogenesis, and tumor growth", Mol Cancer Ther. Diciembre de 2011; 10(12): 2298-308; Bentzien, F., Zuzow, M., Heald, N., Gibson, A., Shi, Y., Goon, L., Yu, P., Engst, S., Zhang, W., Huang, S., Zhao, L., Vysotskaia, V., Chu, F., Bautista, R., Cancilla, B., Lamb, P., Joly, A. and Yakes, M.: "In vitro and in vivo activity of cabozantinib (XL184), an inhibitor of RET, MET, and VEGFR2, in a model of medullary thyroid cancer", Thyroid, 2013 (23) 1569-1577. En los estudios preclínicos, la inhibición mediada por el compuesto 1 de actividad de quinasa produjo una regresión rápida y resistente de vasculatura tumoral, redujo la invasividad y metástasis tumorales y prolongó supervivencia. Véanse, Sennino B, Ishiguro-Oonuma T, Wei Y, Naylor RM, Williamson CW, Bhag- wandin V, Tabruyn SP, You WK, Chapman
HA, Christensen JG, Aftab DT, McDonald DM. (2012), "Suppression of tumor invasión and metástasis by concurrent inhibition of c-Met and VEGF signaling in pancreatic neuroendocrine tumors.", Cancer Discovery, 2(3): 270-87 (Publicado electrónicamente, 24/02/2012).
[0014] En otra realización de la divulgación, el compuesto de Fórmula I, la, el compuesto 1 o una sal farmacéuticamente aceptable del mismo, se administra como una composición farmacéutica que comprende un aditivo, diluyente o excipiente farmacéuticamente aceptable.
[0015] En una realización de la divulgación, el compuesto de la Formula I, la o compuesto 1 o una sal farmacéuticamente aceptable del mismo se administra como una composición farmacéutica que comprende un aditivo, diluyente o excipiente farmacéuticamente aceptable para inhibir quinasas de fusión oncogénicas que involucran el receptor de tirosina quinasa huérfano ROS1 que son el resultado de reorganizaciones cromosómicas que llevan a la activación constitutiva de la actividad de quinasa ROS 1. Estas quinasas de fusión ROS1 están dirigidas a la inhibición utilizando el compuesto de la Formula I, la o compuesto 1 o una sal farmacéuticamente aceptable del mismo. Estos compuestos se pueden administrar como una composición farmacéutica que comprende un aditivo, diluyente o excipiente farmacéuticamente aceptable para tratar el adenocarcinoma de pulmón, por ejemplo, carcinoma de células no pequeñas que alberga una o más quinasas de fusión ROS1 oncogénicas.
[0016] En otro aspecto, la divulgación proporciona un procedimiento para inhibir una quinasa ROS 1 y/o actividad de quinasa de fusión de ROS 1 en uno o más cánceres de pulmón de células no pequeñas o líneas celulares NSCLC que comprende administrar una cantidad terapéuticamente eficaz de una composición farmacéutica que comprende un compuesto de la Fórmula I o la sal de malato de un compuesto de la Fórmula I u otra sal farmacéuticamente aceptable de un compuesto de la Fórmula I a dichos cánceres de pulmón de células no pequeñas o líneas celulares de NSCLC que albergan una quinasa ROS1 y/o un polipéptido de fusión ROS 1.
[0017] En otro aspecto, la divulgación proporciona un procedimiento para detectar, diagnosticar y tratar NSCLC positivo en la fusión de ROS1, por ejemplo, un NSCLC positivo en la fusión de SLC34A2-ROS1 y otros NSCLC conocidos positivo en la fusión de ROS1 , que incluyen fusiones de ROS1 en CD74-ROS1, FIG-ROS1, FIG-ROS1(L), FIG-ROS1(S), y FIG-ROS1(VL) y otras proteínas de fusión ROS1 codificadas por el gen de ROS1 en el cromosoma 6 en humanos (Veánse: Bergethon K, Shaw AT, Ou SH, Katayama R, Lovly CM, McDonald NT, Massion PP, Siwak-Tapp C, Gonzalez A, Fang R, Mark EJ, Batten JM, Chen H, Wilner KD, Kwak EL, Clark JW, Carbone DP, Ji H, Engelman JA, Mino-Kenudson M, Pao W, Iafrate AJ; "ROS1 rearrangements define a unique molecular class of lung cancers", J Clin Oncol. 10 de marzo de 2012; 30(8): 863-70) que comprende administrar una cantidad terapéuticamente eficaz de una composición farmacéutica que comprende un compuesto de la Fórmula I u otra sal farmacéuticamente aceptable de un compuesto de la Fórmula I a un paciente que necesita tal tratamiento. En una realización específica de la divulgación, el compuesto de la Fórmula I es un compuesto 1 o la sal de malato del compuesto 1, particularmente, la sal de S-malato del compuesto 1.
[0018] En otro aspecto, la divulgación proporciona un procedimiento para tratar un adenocarcinoma de pulmón que es cáncer de pulmón de células no pequeñas positivo en la fusión de SLC34A2- ROS1 en un paciente que necesita tal tratamiento, que comprende administrar al paciente una cantidad efectiva de compuesto 1:
o una sal farmacéuticamente aceptable del mismo.
[0019] En otro aspecto, la divulgación proporciona un procedimiento para tratar un adenocarcinoma de pulmón que es cáncer de pulmón de células no pequeñas positivo en la fusión de CD74-ROS1 50 en un paciente que necesita tal tratamiento, que comprende administrar al paciente una cantidad efectiva de compuesto 1:
o una sal farmacéuticamente aceptable del mismo.
[0020] En otro aspecto, la divulgación proporciona un procedimiento para tratar un adenocarcinoma de pulmón que es cáncer de pulmón de células no pequeñas positivo en la fusión de FIG-ROS1 en un paciente que necesita tal tratamiento, que comprende administrar al paciente una cantidad efectiva de compuesto 1:
o una sal farmacéuticamente aceptable del mismo.
[0021] En otro aspecto, la divulgación proporciona un procedimiento para inhibir la actividad de quinasa de fusión de ROS1 en una célula de NSCLC, comprendiendo el procedimiento poner en contacto dicha célula con un a cantidad efectiva de un compuesto de la Fórmula I:
o una sal farmacéuticamente aceptable del mismo, donde:
R1 es halo;
R2 es halo;
R3 es alquilo (C1-Ca);
R4 es alquilo (Ci-Ca); y
Q es CH o N.
a
Breve Descripción de las Figuras
[0022]
La Figura 1 muestra una representación gráfica de proteínas de fusión ROS1 que se conoce que existen en muestras tumorales de Carcinoma Pulmonar de Células No Pequeñas (NSCLC). Las reorganizaciones de ROS1 en NSCLC, glioblastoma y colangiocarcinoma. CD74(L)/(S), grupo de diferenciación 74, isotermas largas/cortas; EZR, ezrina; FIG, fusionado en glioblastoma; LRIG3, repeticiones ricas en leucina y dominios similares a inmunoglobulina 3; TK de ROS1, dominio tirosina quinasa de ROS1 con compañeros de fusión; SDC4, sindecán-4; SLC34A2(L)/(S), familia portadora de soluto 34 (fosfato de sodio), miembro 2, isoformas largas/cortas; SDC4, sindecán-4; TPM3, tropomiosina 3. Los números de exones E32, E34, E35 o E36 indican puntos de rotura de ROS1. La caja roja indica el dominio transmembrana retenido en ROS1.
La Figura 2 representa la inhibición de fosforilación de fusión in vitro de SLC34A2-ROS1 en células HCC-78 de NSCLC que se administraron en dosis únicas escaladas del compuesto 1 o 3-[(1R)-1-(2,6-dicloro-3-fluorofenil)etoxi]-5-(1-piperidin-4-ilpirazol-4-il)piridin-2-amina (Crizotinib; XALKORI®).
La Figura 3 es la secuencia de aminoácido de una proteína ROS1 humana de ejemplo indicada en el presente documento como SEQ ID NO: 1.
La Figura 4 es una secuencia de aminoácidos de ejemplo del dominio de quinasa ROS1 humana indicada en el presente documento como SEQ ID NO: 2.
Descripción Detallada de la Invención
Abreviaturas y Definiciones
[0023] Las siguientes abreviaturas y términos tienen significados indicados a lo largo de esta solicitud.
[0024] El símbolo “-“ significa un enlace sencillo, "=" significa un doble enlace.
[0025] Cuando las estructuras químicas se representan o se describen, a menos que se indique lo contrario, se asume que todos los carbonos tienen sustitución de hidrógeno para ajustarse a una valencia de cuatro. Por ejemplo, en la estructura en la parte izquierda del esquema a continuación existen nueve hidrógenos implicados. Los nueve hidrógenos se representan en la parte derecha del esquema. A veces, un átomo particular en una estructura se describe con fórmula textual por tener un hidrógeno o hidrógenos como sustitución (hidrógeno definido expresamente), por ejemplo, -CH2CH2-. Un experto en la técnica entiende que las técnicas descriptivas mencionadas anteriormente son comunes en las técnicas químicas para proporcionar brevedad y simplicidad a la descripción de estructuras de todos modos complejos.
entonces, a menos que se defina lo contrario, un sustituyente “R” puede residir sobre cualquier átomo del sistema de anillos, asumiendo la sustitución de un hidrógeno representado, implicado o definido expresamente de uno de los átomos de anillo, siempre y cuando se forme una estructura estable.
[0027] Si un grupo "R" es representado como flotante en un sistema de anillos fusionado, como, por ejemplo, en las fórmulas:
entonces, a menos que se defina lo contrario, un sustituyeme R puede residir sobre cualquier atomo del sistema de anillos fusionado, asumiendo la sustitución de un hidrógeno representado (por ejemplo, el -NH- en la formula anterior), el hidrógeno implicado (por ejemplo, como en la fórmula anterior, donde los hidrógenos no se muestran, pero se entiende que están presentes) o hidrógeno definido expresamente (por ejemplo, donde en la fórmula anterior, “Z” es igual a =CH-) de uno de los átomos de anillo, siempre y cuando se forme una estructura estable. En el ejemplo representado, el grupo “R” puede residir o sobre el anillo de 5 miembros o sobre el de 6 miembros del sistema de anillos fusionado. Si un grupo "R" está representado como existente en un sistema de anillos que contiene carbonos saturados, como, por ejemplo, en la fórmula:
donde, en este ejemplo, “y” puede ser más de uno, asumiendo que cada uno sustituye un hidrógeno representado, implicado o definido expresamente en el anillo; entonces, a menos que se defina lo contrario, cuando la estructura resultante es estable, dos “Rs” pueden residir sobre el mismo carbono. Un ejemplo sencillo es cuando R es un grupo metilo; puede existir un grupo dimetilo geminal sobre un carbono del anillo representado (un carbono “anular”). En otro ejemplo, dos Rs en el mismo carbono, incluyendo ese carbono, pueden formar un anillo, creando así una estructura de anillo espirocíclico (un grupo “espirocíclico”) con el anillo representado, como, por ejemplo, en la fórmula:
[0028] "Halógeno" o "halo" se refiere a flúor, cloro, bromo o yodo.
[0029] Tal como se usa en el presente documento, proteína o polipéptido "ROS1" incluye ROS1 de mamífero, por ejemplo, proteína quinasa ROS1 humana (codificada por el gen de ROS1) que es un receptor tirosina quinasa de 2347 aminoácidos de largo que es propenso a una expresión aberrante que provoca cáncer. Una descripción de la quinasa ROS1 humana de longitud completa (con la secuencia de aminoácidos de la proteína ROS1 humana) se puede encontrar en UniProt Número de Adhesión P08922. Adicionalmente, existen múltiples variantes de origen natural de ROS1 (véanse, por ejemplo, Greenman et al., Nature 446: 153-158, 2007). Se conocen las secuencias de nucleótidos y aminoácidos de ROS1 murina de longitud completa (ver, por ejemplo, UniProt Número de Adhesión Q78DX7). Utilizando experimentación rutinaria, el biólogo experto sería fácilmente capaz de determinar las secuencias correspondientes en homólogos de ROS1 de mamífero no humano.
[0030] Por ROS1 de “tipo salvaje” se entiende la quinasa ROS1 de longitud completa (es decir, para ROS1 humana, el polipéptido de 2347 aminoácidos de largo (SEQ ID NO:1) o polipéptido de 2320 aminoácidos de largo (secuencia madura) después de eliminación de la secuencia de péptido señal) en tejido sano (o normal) (por ejemplo, tejido no canceroso) de un individuo normal (por ejemplo, un individuo normal que no sufre de cáncer). No parece que la quinasa ROS1 (de longitud completa o truncada) se exprese en tejido pulmonar normal en humanos. Sin embargo, utilizando los procedimientos descritos a continuación, los inventores han hecho un descubrimiento inesperado de inhibición específica de quinasa ROS1 en células de cáncer de pulmón utilizando los compuestos descritos en el presente documento. Tal expresión en una célula atípica (en este caso una célula cancerosa), donde no se ve ninguna expresión en una célula típica (por ejemplo, una célula pulmonar no cancerosa) es aberrante.
[0031] Se ha descrito la quinasa ROS1 expresada de manera aberrante, en forma de una fusión con otra proteína, por ejemplo, SLC34A2, CD74 o FIG, en glioblastoma (véanse, Charest et al., Genes Chromosomes Cancer 37: 58-71, 2003; Charest et al., Proc. Natl. Acad. Sci. USA 100: 916-921, 2003) en cáncer de hígado (véanse, por ejemplo, Publicación de PCT Número WO2010/093928) en cáncer de conductos biliares (véanse, Gu et al., PlOs one 6 de enero de 2011; 6(1):e15640. doi: 10.1371/journal.pone.0015640) y en adenocarcinoma NSCLC (véanse, Kurtis D. Davies et al., "Identifying and Targeting ROS1 Gene Fusions in Non-Small Cell Lung Cancer", Clin Cancer Res., 1 de septiembre de 2012; 18(17): 4570-4579).
[0032] Tal como se usa en el presente documento, el término “fusión de ROS1” se refiere a una porción del polipéptido ROS1 que comprende el dominio quinasa de la proteína ROS1 (o polinucleótido que codifica el mismo) fusionado a todo o una porción de otro polipéptido (o polinucleótido que codifica el mismo), donde el nombre de este segundo polipéptido o polinucleótido se nombra en la fusión. El término “fusión” significa simplemente todo o una porción de un polipéptido o polinucleótido del primer gen fusionado a todo o una porción de un polipéptido o polinucleótido de un segundo gen. Por ejemplo, una fusión de SLC34A2-ROS1 es una fusión entre una porción del polipéptido SLC34A2 (o polinucleótido que codifica el mismo) y una porción del polipéptido ROS1 (o polinucleótido que codifica el mismo) que comprende el domino quinasa ROS1. Una fusión de ROS1 a menudo es el resultado de una translocación o una inversión cromosómica. Existen numerosas fusiones de ROS1 conocidas, todas ellas son fusiones de ROS1 de la divulgación e incluyen, sin limitación, las proteínas de fusión de SLC34A2-ROS1, cuyos miembros incluyen SLC34A2-ROS1(VS), SLC34A2-ROS1(S), SLC34A2-ROS1(L) (ver, Publicación de Patente de Estados Unidos Número 2010/0143918), CD74-ROS1 (ver, Publicación de Patente de Estados Unidos Número 2010/0221737) y las proteínas de fusión de FIG-ROS1, cuyos miembros incluyen FIG-ROS1(S), FIG-ROS1(L) y FIG-ROS1(XL) (ver, Publicación PCT Número WO2010/093928). La denominación de (L), (S), (v S), (VL) (XS) o (Xl ) después de un polipéptido ROS1 o una fusión de ROS1 se refiere generalmente a polipéptido o polinucleótido ROS1 largo, corto, muy corto, muy largo, extracorto y extralargo, respectivamente.
[0033] Todas las proteínas de fusión de ROS1 conocidas comprenden el domino quinasa completo de ROS1 de longitud completa. Por lo tanto, tal como se usa en el presente documento, por un “polipéptido con actividad de quinasa ROS1” (o “polipéptido que tiene actividad de quinasa ROS1”) se entiende una proteína (o polipéptido) que incluye el dominio quinasa completo de proteína ROS1 de longitud completa y, por lo tanto, retiene la actividad de quinasa ROS1. Los ejemplos no limitantes de proteínas con actividad de quinasa ROS1 incluyen, sin limitación, proteína ROS 1 de longitud completa, las proteínas de fusión de SLC34A2-ROS1, cuyos miembros incluyen SLC34A2-ROS1(VS), SLC34A2-ROS1(S), SLC34A2- ROS1(L) (véase, Publicación de Patente de Estados Unidos Número 2010/0143918), CD74-ROS1 (véase, Publicación de Patente de Estados Unidos Número 2010/0221737) y las proteínas de fusión de FIG-ROS1, cuyos miembros incluyen FIG-ROS1(S), FIG-ROS1(L) y FIG-ROS1(XL) (véase, Publicación PCT Número WO2010/093928) y cualquier forma truncada o mutada de quinasa ROS1 que retiene el dominio quinasa de proteína quinasa ROS1 de longitud completa. Un dominio quinasa de ejemplo de ROS1 se expone en SEQ ID NO: 2, un “polipéptido con actividad de quinasa ROS1” es uno cuya secuencia de aminoácidos comprende SEQ ID NO: 2, o una porción activa de quinasa de la misma.
[0034] Tal como se usa en el presente documento, "polipéptido" (o “secuencia de aminoácidos” o "proteína") se refiere a un polímero formado a partir del enlace, con un orden definido, de, preferiblemente, a-aminoácidos, D-, L-aminoácidos y combinaciones de los mismos. Se hace referencia al enlace entre un residuo de aminoácido y el siguiente como un enlace amida o un enlace peptídico. Los ejemplos no limitantes de polipéptidos incluyen referencias a una secuencia de oligopéptido, péptido, polipéptido o proteína y fragmentos o porciones de la misma y a moléculas de origen natural o sintéticas. Los polipéptidos incluyen, también, moléculas derivatizadas, tales como, glicoproteínas y lipoproteínas, así como, polipéptidos con peso molecular más bajo. “Secuencia de aminoácidos” y los términos similares, tales como, “polipéptido” o “proteína” no intentan limitar la secuencia de aminoácidos indicada a la secuencia de aminoácidos nativa completa asociada con la molécula de proteína mencionada.
[0035] Se entenderá en la técnica que algunas secuencias de aminoácidos de un polipéptido de la divulgación se pueden variar sin efecto significativo de la estructura o la función de la proteína mutante. Si se consideran tales diferencias en la secuencia, se debe recordar que existirán áreas críticas en la proteína que determinan la actividad (por ejemplo, el dominio quinasa de ROS1). En general, es posible reemplazar los residuos que forman la estructura terciaria, siempre que se usen los residuos que realizan una función similar. En otros ejemplos, el tipo de residuo puede ser completamente insignificante si la alteración se produce en una región no crítica de la proteína.
[0036] Por lo tanto, un polipéptido con actividad ROS1 de la presente divulgación incluye además variantes de la proteína ROS1 de longitud completa o varios polipéptidos de fusión de ROS1 descritos en el presente documento que muestran actividad sustancial de quinasa ROS1. Algunas sustituciones conservadoras no limitativas incluyen el intercambio, uno por otro, entre los aminoácidos alifáticos Ala, Val, Leu y Ile; intercambio de los residuos de hidroxilo Ser y Thr; intercambio de los residuos ácidos Asp y Glu; intercambio de los residuos de amida Asn y Gln; intercambio de los residuos básicos Lys y Arg; e intercambio de los residuos aromáticos Phe y Tyr. Otros ejemplos de sustituciones conservadoras de aminoácido conocidos para los expertos en la técnica son: Aromáticos: fenilalanina, triptófano, tirosina (por ejemplo, un residuo de triptófano se sustituye por una fenilalanina); Hidrofóbicos: leucina, isoleucina, valina; Polares: glutamina, asparagina; Básicos: arginina, lisina, histidina; Ácidos: ácido aspártico, ácido glutámico; Pequeños: alanina, serina, treonina, metionina, glicina. Tal como se indica en detalle anteriormente, se puede encontrar una guía adicional en Bowie et al., Science 247, supra con respecto a cuál de los cambios de aminoácidos es probablemente fenotípicamente silencioso (es decir, no se espera que tenga un efecto nocivo sobre una función).
[0037] "SLC34A2" puede hacer referencia a proteína 2B de transporte de fosfato dependiente de sodio codificada por la proteína del gen de SLC34A2 humano o el gen de SLC34A2. La proteína humana SLC34A2 (isoforma A, que tiene un número de Adhesión de NCBI NP_001171469) es una proteína de 689 aminoácidos. SLC34A2 se describe más detalladamente a continuación en el presente documento.
[0038] CD74 se refiere también como la cadena gamma de antígeno de histocompatibilidad de HLA de clase II, también conocida como cadena invariable asociada a antígenos de HLA-DR o CD74 (Grupo de Diferenciación 74), es una proteína que en los humanos es codificada por el gen de CD74. Una proteína CD74 humana ilustrativa tiene un número de adhesión de NCBI NP_001020329 y un número de adhesión de ARNm NM_001025158. CD74 tiene 160 aminoácidos y es codificada por el gen CD74 ubicado en el cromosoma 5 (5q32). CD74 tiene 9 exones. CD74 se describe más detalladamente a continuación en el presente documento.
[0039] FIG (Fusionado en Glioblastoma) denominado también como "PDZ asociado a Golgi y motivo de bobina enrollada que contiene" o proteína "GOPC", es codificada por un gen ubicado en cromosoma 6 en 6q21. Esta proteína es humana y tiene 462 aminoácidos. FIG tiene 3 isoformas producidas por empalme alternativo. Una proteína FIG humana de ejemplo a la que se hace referencia en la presente divulgación tiene un identificador UniProtKB Q9HD26-1.
[0040] "Proteína o gen de fusión de SLC34A2-ROS1" se refiere a fusiones de genes somáticos de los socios SLC34A2 y ROS1. En algunas realizaciones, la proteína de fusión SLC34A2-ROS1 incluye un dominio N-terminal de un socio de fusión, tal como, SLC34A2 y el dominio quinasa C-terminal de proteína ROS1. El dominio N-terminal de un socio de fusión se puede situar en N-terminal de la proteína de fusión, y el dominio quinasa C-terminal de proteína ROS1 se puede situar en C-terminal de la proteína de fusión. El socio de fusión puede ser un dominio N-terminal de la proteína SLC34A2, que se sitúa en N-terminal de la proteína de fusión. En este caso, la proteína de fusión se puede representar como proteína SLC34A2-ROS1 que incluye dominio N-terminal de la proteína SLC34A2 en N-terminal y dominio quinasa C-terminal de la proteína ROS1 en C-terminal. Otra realización da a conocer un gen de fusión que codifica la proteína de fusión, donde un gen que codifica el dominio N-terminal del socio de fusión se sitúa en extremo 5' y un gen que codifica el dominio quinasa C-terminal de la proteína ROS1 se sitúa en extremo 3'. En algunas realizaciones, las porciones de la proteína SLC34A2 se fusionan en marca con porciones de la proteína ROS1 que comprende un domino quinasa funcional como un resultado de una translocación cromosómica aberrante que produce una reorganización cromosómica de ROS1 con SLC34A2. En algunas realizaciones, la proteína de fusión SLC34A2-ROS1 puede incluir la fusión exón 32 de ROS1 y exón 4 de SLC34A2. En algunas realizaciones, la proteína de fusión SLC34A2-ROS1 puede incluir la proteína o el gen de fusión SLC34A2-ROS1 de exón 34 de ROS1 y exón 4 de SLC34A2 que tiene un domino quinasa ROS1 funcional, tal como se usa en la presente divulgación, puede contener otros puntos de rotura que involucran los dos socios de translocación de SLC34A2 y ROS1 Las proteínas de fusión SLC34A2-ROS1 de ejemplo se pueden encontrar en el Catálogo COSMIC de mutaciones somáticas en la base de datos de cáncer v.68 en cancer.sanger.ac.uk/cancergenome/projects/cosmic/.
[0041] "Proteína o gen de fusión CD74-ROS1" se refiere a fusiones de genes somáticos de los socios CD74 y ROS1. Las proteínas de fusión CD74-ROS1 de ejemplo se pueden encontrar en el Catálogo COSMIC de mutaciones somáticas en la base de datos de cáncer v.68 en cancer.sanger.ac.uk/cancergenome/projects/cosmic/.
[0042] "Proteína o gen de fusión FIG-ROS1" se refiere a fusiones de genes somáticos de los socios FIG y ROS1. En algunas realizaciones de la presente divulgación, una eliminación homocigota intracromosómica de 240 kilobases en el cromosoma 6 humano, en la posición 6q21, es responsable de la formación del locus de FIG-ROS1. En algunas realizaciones de la presente divulgación, la transcripción de FIG-ROS1 es codificada por 7 exones de FIG y 9 exones derivados de ROS1. En algunas realizaciones de la presente divulgación, el gen de FIG-ROS1 codifica para una proteína de fusión en marco con un domino quinasa ROS1 constitutivamente activo y funcional. Las proteínas de fusión FIG-ROS1 de ejemplo se pueden encontrar en el Catálogo COSMIC de mutaciones somáticas en la base de datos de cáncer v.68 en cancer.sanger.ac.uk/cancergenome/projects/cosmic/.
[0043] "Paciente" para los propósitos de la presente divulgación incluye humanos y otros animales, particularmente, mamíferos, y otros organismos. Por tanto, los procedimientos son aplicables tanto para terapia humana como a aplicaciones veterinarias. En una realización de la divulgación, el paciente es un mamífero y en otra realización de la divulgación, el paciente es humano.
[0044] “Sal farmacéuticamente aceptable” de un compuesto significa una sal que es farmacéuticamente aceptable y que posee la actividad farmacológica deseada del compuesto parental. Se entiende que las sales farmacéuticamente aceptables son no tóxicas. Información adicional sobre sales farmacéuticamente aceptables adecuadas se puede encontrar en Remington's Pharmaceutical Sciences, 17a edición, Mack Publishing Company, Easton, PA, 1985, o S. M. Berge, et al., "Pharmaceutical Salts," J. Pharm. Sci., 1977; 66: 1-19.
[0045] Los ejemplos de sales de adición de ácido farmacéuticamente aceptables incluyen aquellas formadas con ácidos inorgánicos, tales como ácido clorhídrico, ácido bromhídrico, ácido sulfúrico, ácido nítrico, ácido fosfórico y similares; así como ácidos orgánicos, tales como ácido acético, ácido trifluoroacético, ácido propiónico, ácido hexanoico, ácido ciclopentanopropiónico, ácido glicólico, ácido pirúvico, ácido láctico, ácido oxálico, ácido maleico, ácido malónico, ácido succínico, ácido fumárico, ácido tartárico, ácido málico, ácido cítrico, ácido benzoico, ácido cinámico, ácido 3-(4-hidroxibenzoil)benzoico, ácido mandélico, ácido metanosulfónico, ácido etanosulfónico, ácido 1,2-etanodisulfónico, ácido 2-hidroxietanosulfónico, ácido bencensulfónico, ácido 4-clorobencenosulfónico, ácido 2-naftalenosulfónico, ácido 4-toluenosulfónico, ácido alcanforsulfónico, ácido glucoheptónico, 4,4'-metilenebis-(ácido 3-hidroxi-2-eno-1-carboxílico), ácido 3-fenilpropiónico, ácido trimetilacético, ácido butilacético terciario, ácido lauril
sulfúrico, ácido glucónico, ácido glutámico, ácido hidroxinaftoico, ácido salicílico, ácido esteárico, ácido mucónico, ácido p-toluenosulfónico y ácido salicílico y los similares.
[0046] "Profármaco" se refiere a compuestos que se transforman (típicamente rápidamente) in vivo para producir el compuesto parental de la fórmula mencionada anteriormente, por ejemplo, mediante hidrólisis en sangre. Los ejemplos habituales incluyen, pero no se limitan a, formas de éster y amida de un compuesto que tiene una forma activa que contiene una fracción de ácido carboxílico. Los ejemplos de ésteres farmacéuticamente aceptables de los compuestos de esta divulgación incluyen, pero no se limitan a, ésteres de alquilo (por ejemplo, con entre aproximadamente uno y aproximadamente seis carbonos), el grupo alquilo es una cadena lineal o ramificada. Los ésteres aceptables incluyen también ésteres de cicloalquilo y ésteres de arilalquilo, tales como, pero no se limitan a, bencilo. Los ejemplos de amidas farmacéuticamente aceptables de los compuestos de esta divulgación incluyen, pero no se limitan a, amidas primarias y amidas de alquilo secundarias y terciarias (por ejemplo, con entre aproximadamente uno y aproximadamente seis carbonos). Las amidas y los ésteres de los compuestos de la presente divulgación se pueden preparar según procedimientos convencionales. Se da a conocer una discusión detallada de profármacos en T. Higuchi y V. Stella, "Pro-drugs as Novel Delivery Systems," Volumen 14 de the A.C.S. Symposium Series, y en Bioreversible Carriers in Drug Design, edición Edward B. Roche, American Pharmaceutical Association y Pergamon Press, 1987.
[0047] "ROS1" o la "proteína ROS1" es un receptor transmembrana de tirosina quinasa y se describe más detalladamente en el presente documento.
[0048] "Cantidad terapéuticamente eficaz" es una cantidad de un compuesto de la presente divulgación, que cuando se administra a un paciente, alivia un síntoma de la enfermedad. Una cantidad terapéuticamente eficaz pretende incluir una cantidad de un compuesto solo o en combinación con otros ingredientes activos eficaces para modular c-Met y/o VEGFR2 o eficaz para tratar o prevenir cáncer. La cantidad de un compuesto de la presente divulgación que constituye una “cantidad terapéuticamente eficaz” variará dependiendo del compuesto, el estado de enfermedad y su gravedad, la edad del paciente a tratar y similares. La cantidad terapéuticamente eficaz se puede determinar por un experto en la técnica en vista de su conocimiento y de esta divulgación.
[0049] "Tratar" o "tratamiento" de una enfermedad, trastorno o síndrome, tal como se usa en el presente documento, incluye (i) prevenir la enfermedad, trastorno o síndrome en un ser humano, es decir, causar que los síntomas clínicos de la enfermedad, trastorno o síndrome no se desarrollen en un animal que puede estar expuesto a o predispuesto a la enfermedad, trastorno o síndrome, pero que aun no ha experimentado o mostrado síntomas de la enfermedad, trastorno o síndrome; (ii) revertir o inhibir la enfermedad, trastorno o síndrome, es decir, detener su desarrollo; y (iii) aliviar la enfermedad, trastorno o síndrome, es decir, causar la regresión de la enfermedad, trastorno o síndrome. Tal como se conoce en la técnica, los ajustes de administración sistemática en comparación con localizada, edad, peso corporal, salud general, sexo, dieta, tiempo de administración, interacción del fármaco y la gravedad de la afección pueden ser necesarios, y serán verificables mediante experiencia rutinaria.
[0050] "Producción" para cada una de las reacciones descritas en el presente documento se expresa como un porcentaje de la producción teorética.
Realizaciones
[0051] En una realización de la presente divulgación, el compuesto de la Fórmula I es un compuesto de Fórmula Ia:
o una sal farmacéuticamente aceptable del mismo, donde:
R1 es halo;
R2 es halo; y
Q es CH o N.
[0052] En otra realización de la presente divulgación, el compuesto de la Fórmula I es compuesto 1:
o una sal farmacéuticamente aceptable del mismo. Tal como se ha indicado previamente, al compuesto 1 se hace referencia en el presente documento como N-(4-{[6,7- bis(metiloxi)quinolin-4-il]oxi}fenil)-N'-(4-fluorofenil)ciclopropano-1,1-dicarboxamida. WO 2005/030140 da a conocer el compuesto 1 y describe como se produce (WO 2005/030140, Ejemplos 12, 37, 38 y 48) y, también, da a conocer la actividad terapéutica de este compuesto para inhibir, regular y/o modular la transducción de señales de quinasas, (WO 2005/030140, Ensayos, Tabla 4, entrada 289). El ejemplo 48 está en el párrafo [0353] en el documento WO 2005/030140.
[0053] En otras realizaciones de la presente divulgación, el compuesto de Fórmula I, la, el compuesto 1 o una sal farmacéuticamente aceptable de los mismos se administran como una composición farmacéutica, donde la composición farmacéutica comprende un portador, excipiente o diluyente farmacéuticamente aceptable. En una realización especifica de la presente divulgación, el compuesto de la Fórmula I es el compuesto 1.
[0054] El compuesto de la Fórmula I, Fórmula Ia y compuesto I, tal como se describen en el presente documento, incluye los compuestos mencionados e isómeros individuales y mezclas de isómeros. En cada ejemplo, el compuesto de la Fórmula I incluye las sales, hidratos y/o solvatos farmacéuticamente aceptables de los compuestos mencionados y cualquier isómero individual o mezclas de isómeros de los mismos.
[0055] En otras realizaciones de la divulgación, el compuesto de la Fórmula I, Ia o compuesto 1 pueden ser la sal de (L)-malato. La sal de malato del compuesto de la Fórmula I y del compuesto 1 se da a conocer en PCT/US2010/021194 y USSN 61/325095.
[0056] En otras realizaciones de la divulgación, el compuesto de la Fórmula I es la sal de (D)-malato, a la que también se refiere como la sal de R-malato.
[0057] En otras realizaciones de la divulgación, el compuesto de la Fórmula I es la sal de malato.
[0058] En otras realizaciones de la divulgación, el compuesto de la Fórmula I es la sal de (L)-malato, a la que también se refiere como la sal de S-malato.
[0059] En otras realizaciones de la divulgación, el compuesto 1 es la sal de (D)-malato, a la que también se hace referencia como la sal de S-malato.
[0060] En otras realizaciones de la divulgación, el compuesto 1 es la sal de malato.
[0061] En otras realizaciones de la divulgación, el compuesto 1 es la sal de (L)-malato, a la que también se refiere como la sal de S-malato.
[0062] En otra realización de la divulgación, la sal de malato es la forma cristalina N-1 o la forma N-2 de la sal de (L) malato y/o la sal de (D) malato del compuesto 1, tal como se describe en la Solicitud de Patente de Estados Unidos Número de Serie 61/325095. Véase, también, WO 2008/083319 para las propiedades de enantiómeros cristalinos, que incluyen las formas cristalinas de N-1 y/o N-2, de la sal de malato del compuesto 1. Los procedimientos de producción y caracterización de tales formas se describen detalladamente en PCT/US10/021194.
[0063] En otra realización, la presente divulgación está dirigida a un procedimiento para invertir o inhibir NSCLC, que comprende administrar a un paciente, que necesita tal tratamiento, una cantidad terapéuticamente eficaz de un compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo en cualquiera de las realizaciones descritas en el presente documento. En una realización especifica de la presente divulgación, el compuesto de la Fórmula I es el compuesto 1 o una sal farmacéuticamente aceptable del mismo.
[0064] En otra realización, la presente divulgación está dirigida a un procedimiento para invertir o inhibir NSCLC positivo de la fusión SLC34A2- ROS1, que comprende administrar a un paciente, que necesita tal tratamiento, una cantidad terapéuticamente eficaz de un compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo en cualquiera de las realizaciones descritas en el presente documento. En una realización específica de la presente divulgación, el compuesto de la Fórmula I es el compuesto 1 o una sal farmacéuticamente aceptable del mismo.
[0065] En otra realización, la presente divulgación está dirigida a un procedimiento para invertir o inhibir NSCLC positivo de la fusión CD74- ROS1, que comprende administrar a un paciente, que necesita tal tratamiento, una cantidad terapéuticamente eficaz de un compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo en cualquiera de las realizaciones descritas en el presente documento. En una realización específica de la presente divulgación, el compuesto de la Fórmula I es el compuesto 1 o una sal farmacéuticamente aceptable del mismo.
[0066] En otra realización, la presente divulgación está dirigida a un procedimiento para invertir o inhibir NSCLC positivo de la fusión FIG-ROS1, que comprende administrar a un paciente, que necesita tal tratamiento, una cantidad terapéuticamente eficaz de un compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo en cualquiera de las realizaciones descritas en el presente documento. En una realización específica de la presente divulgación, el compuesto de la Fórmula I es el compuesto 1 o una sal farmacéuticamente aceptable del mismo.
[0067] En otra realización de la presente divulgación, el compuesto de la Fórmula I, o una sal farmacéuticamente aceptable del mismo se administra antes, simultáneamente o posteriormente a uno o más tratamientos. En otra realización, el compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo se administra posteriormente a uno o más tratamientos. “Tratamiento” significa cualquiera de las opciones de tratamiento disponibles para el experto, que incluyen cirugía, agentes quimioterapéuticos, terapias hormonales, inmunoterapias, terapia con yodo radioactivo y radiación. En particular, “tratamiento” significa otro agente quimioterapéutico o anticuerpo.
[0068] Por lo tanto, en otra realización de la presente divulgación, el compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con cisplatino y/o gemcitabina.
[0069] En otra realización de la presente divulgación, el compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con docetaxel.
[0070] En otra realización de la presente divulgación, el compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con anticuerpos HER-2. En otra realización, el anticuerpo HER-2 es trastuzumab.
[0071] En otra realización de la presente divulgación, el compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con cisplatino y/o gemcitabina y/o docetaxel.
[0072] En otra realización de la presente divulgación, el compuesto de la Fórmula I, Ia o compuesto 1 o una sal farmacéuticamente aceptable del mismo se administra por vía oral una vez al día como un comprimido o una cápsula. En estas y otras realizaciones, el compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo es el compuesto 1 o una sal farmacéuticamente aceptable del mismo.
[0073] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral como su base libre o la sal de malato como una cápsula o un comprimido.
[0074] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral una vez al día como su base libre o como la sal de malato como una cápsula o un comprimido que contiene hasta 100 mg del compuesto 1.
[0075] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral una vez al día como su base libre o como la sal de malato como una cápsula o un comprimido que contiene 100 mg del compuesto 1.
[0076] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral una vez al día como su base libre o como la sal de malato como una cápsula o un comprimido que contiene 95 mg del compuesto 1.
[0077] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral una vez al día como su base libre o como la sal de malato como una cápsula o un comprimido que contiene 90 mg del compuesto 1.
[0078] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral una vez al día como su base libre o como la sal de malato como una cápsula o un comprimido que contiene 85 mg del compuesto 1.
[0079] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral una vez al día como su base libre o como la sal de malato como una cápsula o un comprimido que contiene 80 mg del compuesto 1.
[0080] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral una vez al día como su base libre o como la sal de malato como una cápsula o un comprimido que contiene 75 mg del compuesto 1.
[0081] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral una vez al día como su base libre o como la sal de malato como una cápsula o un comprimido que contiene 70 mg del compuesto 1.
[0082] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral una vez al día como su base libre o como la sal de malato como una cápsula o un comprimido que contiene 65 mg del compuesto 1.
[0083] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral una vez al día como su base libre o como la sal de malato como una cápsula o un comprimido que contiene 60 mg del compuesto 1.
[0084] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral una vez al día como su base libre o como la sal de malato como una cápsula o un comprimido que contiene 55 mg del compuesto 1.
[0085] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral una vez al día como su base libre o como la sal de malato como una cápsula o un comprimido que contiene 50 mg del compuesto 1.
[0086] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral una vez al día como su base libre o como la sal de malato como una cápsula o un comprimido que contiene 45 mg del compuesto 1.
[0087] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral una vez al día como su base libre o como la sal de malato como una cápsula o un comprimido que contiene 40 mg del compuesto 1.
[0088] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral una vez al día como su base libre o como la sal de malato como una cápsula o un comprimido que contiene 35 mg del compuesto 1.
[0089] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral una vez al día como su base libre o como la sal de malato como una cápsula o un comprimido que contiene 30 mg del compuesto 1.
[0090] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral una vez al día como su base libre o como la sal de malato como una cápsula o un comprimido que contiene 25 mg del compuesto 1.
[0091] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral una vez al día como su base libre o como la sal de malato como una cápsula o un comprimido que contiene 20 mg del compuesto 1.
[0092] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral una vez al día como su base libre o como la sal de malato como una cápsula o un comprimido que contiene 15 mg del compuesto 1.
[0093] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral una vez al día como su base libre o como la sal de malato como una cápsula o un comprimido que contiene 10 mg del compuesto 1.
[0094] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral una vez al día como su base libre o como la sal de malato como una cápsula o un comprimido que contiene 5 mg del compuesto 1.
[0095] En otra realización de la presente divulgación, el compuesto 1 se administra como su base libre o la sal de malato por vía oral una vez al día como un comprimido, tal como se muestra en la tabla a continuación.
[0096] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral como su base libre o la sal de malato una vez al día como un comprimido, tal como se muestra en la tabla a continuación.
[0097] En otra realización de la presente divulgación, el compuesto 1 se administra por vía oral como su base libre o la sal de malato una vez al día como un comprimido, tal como se muestra en la tabla a continuación.
[0098] Se puede ajustar cualquiera de las formulaciones de comprimidos proporcionados anteriormente a la dosis deseada del compuesto 1 o una sal farmacéuticamente aceptable del mismo. Por consiguiente, se puede ajustar proporcionalmente la cantidad de cada uno de los ingredientes de formulación para proporcionar una formulación de comprimido que contiene varias cantidades del compuesto 1, tal como se proporciona en los parágrafos anteriores. En otra realización de la presente divulgación, las formulaciones pueden contener 20, 40, 60 o 80 mg de compuesto 1 o una sal farmacéuticamente aceptable del mismo.
[0099] En algunas realizaciones, la presente divulgación da a conocer un procedimiento para inhibir o revertir el progreso del crecimiento celular anormal en un mamífero, que comprende administrar un compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo, donde el crecimiento celular anormal es cáncer mediado por una proteína de fusión de ROS1, por ejemplo, proteína de fusión SLC34A2-ROS1, proteína de fusión CD74-ROS1, proteína de fusión FIG-ROS1, proteína de fusión FIG-ROS1(L), proteína de fusión FIG-ROS1(S) y proteína de fusión FIG-ROS1(VL) y otras proteínas de fusión de ROS1, que comprenden un dominio quinasa de ROS1 C-terminal funcional como el que se codifica por el gen de ROS1 en el cromosoma 6 en humanos (6q22). En una realización de la presente divulgación, el cáncer es adenocarcinoma de pulmón. En otra, el adenocarcinoma de pulmón es cáncer de pulmón de células no pequeñas. En otra realización de la presente divulgación, el adenocarcinoma de pulmón es cáncer de pulmón de células no pequeñas positivo en la fusión de SLC34A2-ROS1. En otra realización de la presente divulgación, el adenocarcinoma de pulmón es cáncer de pulmón de células no pequeñas positivo en la fusión de CD74-ROS1. En otra realización de la presente divulgación, el adenocarcinoma de pulmón es cáncer de pulmón de células no pequeñas positivo en la fusión de FIG-ROS1. En otra realización de la presente divulgación, el adenocarcinoma de pulmón es cáncer de pulmón de células no pequeñas positivo en la fusión de ROS1. En otra realización de la presente divulgación, un compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo se administra como una composición farmacéutica que comprende un compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo y al menos un portador farmacéuticamente aceptable. En otra realización de la presente divulgación, el
compuesto de la Fórmula I se administra después de otra forma de tratamiento. En otra realización de la presente divulgación, un compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con cisplatino y/o gemcitabina. En otra realización de la presente divulgación, un compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con carboplatino y/o gemcitabina. En otra realización de la presente divulgación, un compuesto de la Fórmula I se administra después de tratamiento con crizotinib. En otra realización de la presente divulgación, un compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con crizotinib y/o gemcitabina. En otra realización de la presente divulgación, un compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con docetaxel. En otra realización de la presente divulgación, un compuesto de la Fórmula I se administra después de tratamiento con carboplatino. En otra realización de la presente divulgación, un compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con cisplatino y/o gemcitabina y/o docetaxel. En otra realización de la presente divulgación, un compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con carboplatino y/o gemcitabina y/o docetaxel. En otra realización de la presente divulgación, un compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con crizotinib y/o gemcitabina y/o docetaxel.
[0100] En algunas realizaciones, la presente divulgación da a conocer un procedimiento para inhibir o revertir el progreso de crecimiento celular anormal en un mamífero, que comprende administrar un compuesto de la Fórmula Ia o una sal farmacéuticamente aceptable del mismo, donde el crecimiento celular anormal es cáncer mediado por una proteína de fusión de ROS1, por ejemplo, proteína de fusión SLC34A2-ROS1, proteína de fusión CD74-ROS1, proteína de fusión FIG-ROS1, proteína de fusión FIG-ROS1(L), proteína de fusión FIG-ROS1(S) y proteína de fusión FIG-ROS1(VL) y otras proteínas de fusión de ROS1, que comprenden un dominio quinasa de ROS1 C-terminal funcional como el que se codifica por el gen de ROS1 en el cromosoma 6 en humanos (6q22). En una realización de la presente divulgación, el cáncer es adenocarcinoma de pulmón. En otra, el adenocarcinoma de pulmón es cáncer de pulmón de células no pequeñas. En otra realización de la presente divulgación, el adenocarcinoma de pulmón es cáncer de pulmón de células no pequeñas positivo en la fusión de SLC34A2-ROS1. En otra realización de la presente divulgación, el adenocarcinoma de pulmón es cáncer de pulmón de células no pequeñas positivo en la fusión de CD74-ROS1. En otra realización de la presente divulgación, el adenocarcinoma de pulmón es cáncer de pulmón de células no pequeñas positivo en la fusión de FIG-ROS1. En otra realización de la presente divulgación, el adenocarcinoma de pulmón es cáncer de pulmón de células no pequeñas positivo en la fusión de ROS1. En otra realización de la presente divulgación, un compuesto de la Fórmula Ia o una sal farmacéuticamente aceptable del mismo se administra como una composición farmacéutica que comprende un compuesto de la Fórmula Ia o una sal farmacéuticamente aceptable del mismo y al menos un portador farmacéuticamente aceptable. En otra realización de la presente divulgación, el compuesto de la Fórmula Ia se administra después de otra forma de tratamiento. En otra realización, un compuesto de la Fórmula Ia o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con cisplatino y/o gemcitabina. En otra realización de la presente divulgación, un compuesto de la Fórmula Ia o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con docetaxel. En otra realización de la presente divulgación, un compuesto de la Fórmula Ia o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con cisplatino y/o gemcitabina y/o docetaxel. En otra realización de la presente divulgación, un compuesto de la Fórmula Ia o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con carboplatino y/o gemcitabina. En otra realización de la presente divulgación, un compuesto de la Fórmula Ia o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con crizotinib. En otra realización de la presente divulgación, un compuesto de la Fórmula Ia o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con crizotinib y/o gemcitabina. En otra realización de la presente divulgación, un compuesto de la Fórmula Ia o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con docetaxel. En otra realización de la presente divulgación, un compuesto de la Fórmula Ia se administra después de tratamiento con carboplatino. En otra realización de la presente divulgación, un compuesto de la Fórmula Ia o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con cisplatino y/o gemcitabina y/o docetaxel. En otra realización de la presente divulgación, un compuesto de la Fórmula Ia o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con carboplatino y/o gemcitabina y/o docetaxel. En otra realización de la presente divulgación, un compuesto de la Fórmula Ia o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con crizotinib y/o gemcitabina y/o docetaxel.
[0101] En otras realizaciones, la presente divulgación da a conocer un procedimiento para inhibir o revertir el progreso de crecimiento celular anormal en un mamífero, que comprende administrar el compuesto 1 o una sal farmacéuticamente aceptable del mismo, donde el crecimiento celular anormal es cáncer mediado por una proteína de fusión de ROS1, por ejemplo, proteína de fusión SLC34A2-ROS1, proteína de fusión CD74-ROS1, proteína de fusión FIG-ROS1, proteína de fusión FIG-ROS1(L), proteína de fusión FIG-ROS1(S) y proteína de fusión FIG-ROS1(VL) y otras proteínas de fusión de ROS1, que comprenden un dominio quinasa de ROS1 C-terminal funcional como el que se codifica por el gen de ROS1 en el cromosoma 6 en humanos (6q22). En una realización de la presente divulgación, el cáncer es adenocarcinoma de pulmón. En otra, el adenocarcinoma de pulmón es cáncer de pulmón de células no pequeñas. En otra realización de la presente divulgación, el adenocarcinoma de pulmón es cáncer de pulmón de células no pequeñas positivo en la fusión de SLC34A2-ROS1. En otra realización de la presente divulgación, el adenocarcinoma de pulmón es cáncer de pulmón de células no pequeñas positivo en la fusión de CD74ROS1. En otra realización, el adenocarcinoma de pulmón es cáncer de pulmón de células no pequeñas positivo en la fusión de FIG-ROS1. En otra realización de la presente divulgación, el adenocarcinoma de pulmón es cáncer de pulmón de células no pequeñas positivo en la fusión de ROS1. En otra realización de la presente divulgación, el compuesto 1 o una sal farmacéuticamente aceptable del mismo se administra como una composición farmacéutica que comprende compuesto 1 o una sal farmacéuticamente aceptable del mismo y al menos un portador farmacéuticamente aceptable. En otra realización de la presente divulgación, el compuesto 1 se administra después de otra forma de tratamiento. En otra realización de la presente divulgación, el compuesto 1 o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con cisplatino y/o gemcitabina. En otra realización de la presente divulgación, el compuesto 1 o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con docetaxel. En otra realización de la presente divulgación, el compuesto 1 o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con cisplatino y/o gemcitabina y/o docetaxel. En otra realización de la presente divulgación, el compuesto 1 o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con carboplatino y/o gemcitabina. En otra realización de la presente divulgación, el compuesto 1 o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con crizotinib. En otra realización de la presente divulgación, el compuesto 1 o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con crizotinib y/o gemcitabina. En otra realización de la presente divulgación, el compuesto 1 o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con docetaxel. En otra realización de la presente divulgación, el compuesto 1 o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con carboplatino. En otra realización de la presente divulgación, el compuesto 1 o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con cisplatino y/o gemcitabina y/o docetaxel. En otra realización de la presente divulgación, el compuesto 1 o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con carboplatino y/o gemcitabina y/o docetaxel. En otra realización de la presente divulgación, el compuesto 1 o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con crizotinib y/o gemcitabina y/o docetaxel. En otra realización de la presente divulgación, el compuesto 1 o una sal farmacéuticamente aceptable del mismo se administra después del tratamiento con crizotinib y/o carboplatino.
[0102] En otras realizaciones, la presente divulgación da a conocer un procedimiento para prevenir, tratar, inhibir o revertir el progreso de crecimiento celular anormal en un mamífero, que comprende administrar el compuesto 1 o una sal farmacéuticamente aceptable del mismo, donde el crecimiento celular anormal es un cáncer mediado por una proteína de fusión de ROS1, por ejemplo, proteína de fusión SLC34A2-ROS1, proteína de fusión CD74-ROS1, proteína de fusión FIG-ROS1, proteína de fusión FIG-ROS1(L), proteína de fusión FIG-ROS1(S), proteína de fusión FIG-ROS1(VL) u otras proteínas de fusión de ROS1, donde el cáncer se trató previamente con un régimen terapéutico de crizotinib y/o carboplatino y es resistente a crizotinib y/o carboplatino. En algunas realizaciones de la presente divulgación, el cáncer que es resistente a crizotinib y/o carboplatino alberga una o más proteínas de fusión de ROS1 seleccionadas de: proteína de fusión SLC34A2-ROS1, proteína de fusión CD74-ROS1, proteína de fusión FIG-ROS1, proteína de fusión FIG-ROS1(L), proteína de fusión FIG-ROS1(S) y proteína de fusión FIG-ROS1(VL) y/o contiene una mutación en el dominio quinasa de ROS1 de la proteína de fusión SLC34A2-ROS1, proteína de fusión CD74-ROS1, proteína de fusión FIG-ROS1, proteína de fusión FIG-ROS1(L), proteína de fusión FIG-ROS1(S) o proteína de fusión FIG-ROS1(VL). En una realización de la presente divulgación, el cáncer resistente a crizotinib y/o carboplatino es un cáncer que tiene una mutación en el dominio quinasa de ROS1 de una proteína de fusión CD74-ROS1. En una realización relacionada de la presente divulgación, el cáncer es un adenocarcinoma de pulmón positivo en la fusión de CD74-ROS1 refractario de crizotinib, por ejemplo, NSCLC positivo en la fusión de CD74-ROS1 refractario de crizotinib que alberga una mutación en el dominio quinasa de ROS1. En otra realización de la presente divulgación, el cáncer resistente a crizotinib y/o carboplatino es un cáncer, por ejemplo, adenocarcinoma de pulmón resistente a crizotinib, por ejemplo, un NSCLC resistente a crizotinib que alberga una mutación seleccionada de E1990G, G2032R, L2026M, L1951r , M2128V, K2003I y las combinaciones de las mismas en el dominio quinasa de ROS1 de una proteína de fusión CD74-ROS1.
[0103] En otra realización, la presente divulgación da a conocer un procedimiento para prevenir, tratar, inhibir o revertir el progreso de crecimiento celular anormal en un mamífero, por ejemplo, un cáncer, comprendiendo el procedimiento administrar un compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo, donde el crecimiento celular anormal es un cáncer mediado por una proteína de fusión de ROS1, por ejemplo, proteína de fusión SLC34A2-ROS1, proteína de fusión CD74-ROS1, proteína de fusión FIG-ROS1, proteína de fusión FIG-ROS1(L), proteína de fusión FIG-ROS1(S), proteína de fusión FIG-ROS1(VL) u otras proteínas de fusión de ROS1, donde el cáncer se ha tratado previamente con un régimen terapéutico de crizotinib y/o carboplatino y es resistente a crizotinib y/o carboplatino. En algunas realizaciones de la presente divulgación, el cáncer que es resistente a crizotinib y/o carboplatino alberga una o más proteínas de fusión de ROS1 seleccionadas de: proteína de fusión SLC34A2-ROS1, proteína de fusión CD74-ROS1, proteína de fusión FIG-ROS1, proteína de fusión FIG- ROS 1(L), proteína de fusión FIG- ROS 1(S) y proteína de fusión FIG-ROS1(VL) y/o contiene una mutación en el dominio quinasa de ROS1 de la proteína de fusión SLC34A2-ROS1, proteína de fusión CD74-ROS1, proteína de fusión FIG-ROS1, proteína de fusión FIG-ROS1(L), proteína de fusión FIG-ROS1(S) o proteína de fusión FIG-ROS1(VL). En una realización de la presente divulgación, el cáncer resistente a crizotinib es un cáncer que tiene una mutación en el dominio quinasa de ROS1 de una proteína de fusión CD74-ROS1. En una realización relacionada de la presente divulgación, el cáncer es un adenocarcinoma de pulmón positivo en la fusión de CD74-ROS1 refractario de crizotinib, por ejemplo, NSCLC positivo en la fusión de CD74-ROS1 refractario de crizotinib que alberga una mutación en el dominio quinasa de ROS1. En
otra realización de la presente divulgación, el cáncer resistente a crizotinib es un cáncer, por ejemplo, un adenocarcinoma de pulmón resistente a crizotinib, por ejemplo, un NSCLC resistente a crizotinib que alberga una mutación seleccionada de E1990G, G2032R, L2026M, L1951R, M2128V, K2003I y las combinaciones de las mismas en el dominio quinasa de ROS1 de una proteína de fusión CD74-ROS1. En diferentes realizaciones de la divulgación descritas anteriormente, un procedimiento para prevenir, tratar, inhibir o revertir el progreso de crecimiento celular anormal en un mamífero comprende administrar un compuesto de la Fórmula I o un compuesto de la Fórmula Ia o un compuesto 1 o una sal farmacéuticamente aceptable de los mismos en una dosis terapéuticamente eficaz de ejemplo que varía de aproximadamente 0,01 mg/kg hasta aproximadamente 100 mg/kg, o de aproximadamente 0,1 mg/kg hasta aproximadamente 75 mg/kg, o de aproximadamente 0,1 mg/kg hasta aproximadamente 50 mg/kg, o de aproximadamente 0,1 mg/kg hasta aproximadamente 40 mg/kg, o de aproximadamente 0,1 mg/kg hasta aproximadamente 30 mg/kg, o de aproximadamente 0,1 mg/kg hasta aproximadamente 20 mg/kg, o de aproximadamente 0,1 mg/kg hasta aproximadamente 15 mg/kg, o de aproximadamente 0,1 mg/kg hasta aproximadamente 10 mg/kg o de aproximadamente 0,1 mg/kg hasta aproximadamente 5 mg/kg, o de aproximadamente 0,1 mg/kg hasta aproximadamente 1 mg/kg a un paciente necesitado, donde el crecimiento celular anormal es un cáncer mediado por una proteína de fusión de ROS1, por ejemplo, proteína de fusión SLC34A2-ROS1, proteína de fusión CD74-ROS1, proteína de fusión FIG-ROS1, proteína de fusión FIG-ROS1(L), proteína de fusión FIG-ROS1(S), proteína de fusión FIG-ROS1(VL) u otra proteína de fusión de ROS1; o un cáncer que alberga una o más mutaciones en una proteína de fusión de ROS1, por ejemplo, una mutación en un dominio quinasa de ROS1 en una proteína de fusión SLC34A2-ROS1, proteína de fusión CD74-ROS1, proteína de fusión FIG-ROS1, proteína de fusión FIG-ROS1(L), proteína de fusión FIG-ROS1(S), proteína de fusión FIG-ROS1(VL) u otras proteínas de fusión ROS1, que incluyen, en algunas realizaciones de la divulgación, un cáncer que ha sido tratado previamente con un régimen terapéutico de crizotinib y/o carboplatino y es resistente a crizotinib y/o carboplatino, por ejemplo, un cáncer resistente a crizotinib, por ejemplo, un adenocarcinoma de pulmón resistente a crizotinib, por ejemplo, un NSCLC resistente a crizotinib.
Adm inistración
[0104] Se puede llevar a cabo la administración del compuesto de Fórmula I, Fórmula la o compuesto 1 o una sal farmacéuticamente aceptable de los mismos, en una forma pura o en una composición farmacéutica utilizando cualquiera de las vías de administración o agentes que se utilizan con fines similares. Por consiguiente, la administración puede ser, por ejemplo, por vía oral, nasal, parenteral (intravenosa, intramuscular o subcutánea), tópica, transdérmica, intravaginal, intravesical, intracistemal o rectal, en forma de sólido, semisólido, polvos liofilizados o formas de administración líquidas, tales como, por ejemplo, comprimidos, supositorios, pastillas, dosis de gelatina blanda elástica y dura (que puede estar en cápsulas o comprimidos), polvos, soluciones, suspensiones o aerosoles o los similares, específicamente en formas de dosificación unitaria adecuadas para administración simple con dosis precisa.
[0105] Las composiciones incluirán un portador o un excipiente farmacéutico convencional y un compuesto de la Fórmula I como el/un agente activo y, adicionalmente, pueden incluir portadores y adyuvantes, etc.
[0106] Los adyuvantes incluyen agentes de conservación, humidificación, suspensión, edulcorantes, saborizantes, aromatizantes, emulsionantes y de administración. La prevención de la acción de microorganismos se puede garantizar utilizando diferentes agentes antibacterianos y antifúngicos, por ejemplo, parabenos, clorobutanol, fenol, ácido sórbico y los similares. También se puede conveniente a incluir agentes isotónicos, por ejemplo, azucares, cloruro de sodio y los similares. El uso de agentes que retrasan absorción, por ejemplo, monoestearato de aluminio y gelatina, puede provocar absorción prolongada de la forma farmacéutica inyectable.
[0107] Si se desea, una composición farmacéutica del compuesto de la Fórmula I puede contener cantidades menores de sustancias auxiliares, tales como, agentes humidificantes o emulsionantes, agentes tampón de pH, antioxidantes y los similares, por ejemplo, ácido cítrico, monolaurato de sorbitán, oleato de trietanolamina; hidroxitolueno butilado, etc.
[0108] La elección de composición depende de varios factores, tales como, el modo de administración de fármaco (por ejemplo, administración oral, composiciones en forma de comprimidos, pastillas o cápsulas) y la biodisponibilidad de material farmacológico. Recientemente, se han desarrollado las composiciones farmacéuticas para fármacos que muestran baja biodisponibilidad basándose en el principio que la biodisponibilidad se puede aumentar aumentando el área de superficie, es decir, disminuyendo el tamaño de partícula. Por ejemplo, la Patente de los Estados Unidos Número 4.107.288 describe una composición farmacéutica que tiene partículas cuyo tamaño oscila desde 10 hasta 1000 nm en las que el material activo se apoya sobre una matriz reticular de macromoléculas. La Patente de los Estados Unidos Número 5.145.684 describe la producción de una composición farmacéutica, donde la sustancia farmacológica se pulveriza sobre nanopartículas (tamaño de partícula medio 400 nm) en presencia de un modificador de superficie y, a continuación, se dispersa en un medio líquido para proporcionar una composición farmacéutica que muestra biodisponibilidad notablemente alta.
[0109] Las composiciones adecuadas para la inyección parenteral pueden comprender soluciones, dispersiones, suspensiones o emulsiones acuosas o no acuosas estériles fisiológicamente aceptables y polvos estériles para rehidratación en soluciones o dispersiones inyectables. Los ejemplos de portadores, diluyentes, disolventes o
vehículos acuosos o no acuosos adecuados incluyen agua, etanol, polioles (propilenglicol, polietilenglicol, glicerol y los similares), mezclas adecuadas de los mismos, aceites vegetales (tal como, aceite de oliva) y ésteres orgánicos inyectables, tal como, oleato de etilo. Se puede mantener fluidez apropiada, por ejemplo, utilizando un recubrimiento, tal como, lecitina, manteniendo el tamaño de partícula requerido en el caso de dispersiones y utilizando tensioactivos.
[0110] Una vía específica de administración es oral, utilizando un régimen de dosis diaria conveniente que se puede ajustar según el nivel de gravedad del estado de enfermedad a tratar.
[0111] Las formas de administración sólidas para administración oral incluyen cápsulas, comprimidos, pastillas, polvos y gránulos. En tales formas de administración, el compuesto activo se incorpora con al menos un excipiente (o portador) habitual inerte, tal como, citrato de sodio o fosfato de dicalcio o (a) rellenos o expansores, tales como, por ejemplo, almidón, lactosa, sacarosa, glucosa, manitol y ácido silícico, (b) aglutinantes, tales como, por ejemplo, derivados de celulosa, almidón, alignatos, gelatina, polivinilpirrolidona, sacarosa y goma arábiga, (c) humectantes, tal como, por ejemplo, glicerol, (d) agentes desintegrantes, tales como, por ejemplo, agar-agar, carbonato de calcio, fécula de patata o tapioca, ácido algínico, croscarmelosa de sodio, silicatos complejos y carbonato de sodio, (e) retardantes de soluciones, tal como, por ejemplo, compuestos de amonio cuaternario, (g) agentes humidificantes, tales como, por ejemplo, alcohol cetílico y monoestearato de glicerol, estearato de magnesio y los similares, (h) absorbentes, tales como, por ejemplo, caolín y bentonita y (i) lubricantes, tales como, por ejemplo, talco, estearato de calcio, estearato de magnesio, glicoles de polietileno sólido, lauril sulfato de sodio o mezclas de los mismos. En el caso de cápsulas, comprimidos y pastillas, las formas de administración pueden comprender también agentes tampón.
[0112] Las dosis de administración sólidas, tales como, descritas anteriormente, se pueden preparar con recubrimientos y capas, tales como, recubrimientos entéricos y otros bien conocidos en la técnica. Pueden contener agentes calmantes y, también, pueden ser de tal composición que liberan el compuesto o los compuestos activo(s) en una parte determinada del tracto intestinal de una manera retardada. Los ejemplos de composiciones integradas que se pueden usar son sustancias poliméricas y ceras. Los compuestos activos, también, pueden ser microencapsulados, si es conveniente, con uno o más excipientes mencionados anteriormente.
[0113] Las formas de administración líquidas para la administración oral incluyen emulsiones, soluciones, suspensiones, siropes y elixires farmacéuticamente aceptables. Tales formas de administración se preparan, por ejemplo, disolviendo, dispersando, etc., el compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo y adyuvantes farmacéuticos opcionales en un portador, tal como, por ejemplo, agua, solución salina, dextrosa acuosa, glicerol, etano y los similares; agentes solubilizantes y emulsionantes, tales como, por ejemplo, alcohol etílico, alcohol isopropílico, carbonato de etilo, acetato de etilo, alcohol bencílico, benzoato de bencilo, propilenglicol, 1,3-butilenglicol, dimetilformamida; aceites, particularmente, aceite de semillas de algodón, aceite de cacahuete, aceite de germen de maíz, aceite de oliva, aceite de ricino y aceite de sésamo, glicerol, alcohol tetrahidrofurfurílico, polietilenglicoles y ésteres de ácidos grasos de sorbitán; o mezclas de estas sustancias y los similares, para producir así una solución o una suspensión.
[0114] Las suspensiones, además de los compuestos activos, pueden contener agentes de suspensión, tales como, por ejemplo, alcoholes etoxilados de isoestearil, sorbitol de polioxietileno y ésteres de sorbitán, celulosa microcristalina, metahidróxido de aluminio, bentonita, agar-agar y tragacanto o mezclas de estas sustancias y los similares.
[0115] Las composiciones para administración rectal son, por ejemplo, supositorios que se pueden preparar mezclando el compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo.con, por ejemplo, excipientes o portadores no irritantes adecuados, tales como, manteca de cacao, polietilenglicol o cera para supositorios, que son sólidos a temperaturas regulares, pero líquidos a temperatura corporal y, por lo tanto, se funden cuando se encuentran en una cavidad corporal adecuada y liberan el componente activo en la misma.
[0116] Las formas de administración para administración tópica del compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo incluyen pomadas, polvos, esprays e inhalantes. El componente activo se incorpora bajo condiciones estériles con un portador fisiológicamente aceptable y cualquier conservante, tampón o propulsor que puedan requerirse. Composiciones oftalmológicas, ungüentos oculares, polvos y soluciones también se contemplan como los que están dentro del ámbito de esta divulgación.
[0117] Se pueden usar gases comprimidos para dispersar el compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo en forma de aerosol. Los gases inertes a tal fin son nitrógeno, dióxido de carbono, etc.
[0118] Generalmente, dependiendo del procedimiento previsto de administración, las composiciones farmacéuticamente aceptables contendrán desde aproximadamente 1% hasta aproximadamente 99% en peso de un compuesto(s) de Fórmula I o una sal farmacéuticamente aceptable del mismo y de 99% hasta 1% en peso de un excipiente farmacéuticamente adecuado. En un ejemplo, la composición será entre aproximadamente 5% y aproximadamente 75% en peso de un compuesto(s) de la Fórmula I, Fórmula Ia o compuesto 1 o una sal farmacéuticamente aceptable de los mismos con el resto siendo excipientes farmacéuticamente adecuados.
[0119] Los procedimientos actuales de preparación de tales formas de administración se conocen o serán evidentes
a aquellos expertos en la técnica; por ejemplo, véanse Remington's Pharmaceutical Sciences, 18a Edición, (Mack Publishing Company, Easton, Pa., 1990). La composición a administrar contendrá, en cualquier caso, una cantidad terapéuticamente eficaz de un compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo para el tratamiento de un estado de enfermedad según las instrucciones de esta divulgación.
[0120] Los compuestos de esta divulgación o sus sales o solvatos farmacéuticamente aceptables se administran en una cantidad terapéuticamente eficaz que variará dependiendo de una variedad de factores que incluyen la actividad del compuesto específico empleado, la estabilidad metabólica y la duración de acción del compuesto, la edad, el peso corporal, la salud general, el sexo, la dieta, el modo y el tiempo de administración, el promedio de excreción, la combinación de fármacos, la gravedad de los estados de enfermedad particulares y la terapia presentada a la que se somete. El compuesto de la Fórmula I, Fórmula la o compuesto 1 o una sal farmacéuticamente aceptable del mismo se puede administrar a un paciente con niveles de dosis que oscilan entre aproximadamente 0,1 y aproximadamente 1,000 mg por día. Para un adulto humano normal que tiene un peso corporal de aproximadamente 70 kilogramos, un ejemplo es una dosis que oscila entre aproximadamente 0,01 y aproximadamente 100 mg por kilogramo de peso corporal. La dosis específica utilizada puede variar. Por ejemplo, la dosis puede depender de un número de factores que incluyen los requisitos del paciente, la gravedad de la condición que se trata y la actividad farmacológica del compuesto que se utiliza. La determinación de las dosis óptimas para un paciente particular se conoce bien por un experto en la técnica.
[0121] En otras realizaciones de la presente divulgación, el compuesto de la Fórmula I, Fórmula la o compuesto 1 o una sal farmacéuticamente aceptable del mismo se administra al paciente de manera simultánea con otros tratamientos de cáncer. Tales tratamientos incluyen otras quimioterapéuticos, terapia de reemplazo hormonal, terapia de radiación o inmunoterapia, entre otros. La elección de otra terapia dependerá de un número de factores que incluyen la estabilidad metabólica y la duración de acción del compuesto, la edad, el peso corporal, la salud general, el sexo, la dieta, el modo y el tiempo de administración, el promedio de excreción, la combinación de fármacos, la gravedad de los estados de enfermedad particulares y la terapia presentada a la que se somete.
[0122] En otro aspecto, la presente divulgación da a conocer un procedimiento para detectar, diagnosticar y tratar enfermedad relacionada con fusiones de ROS1, tales como, NSCLC positivo en la fusión de SLC34A2-ROS1, NSCLC positivo en la fusión de CD74-ROS1 y NSCLC positivo en la fusión de FIG-ROS1, adicionalmente a otros NSCLC positivo en la fusión de ROS1. En el presente documento se describen más detalladamente los procedimientos para detectar y diagnosticar tales trastornos. Se puede conseguir mejor tratamiento de estos trastornos con la administración de una cantidad terapéuticamente eficaz de una composición farmacéutica que comprende un compuesto de la Fórmula I u otra sal farmacéuticamente aceptable de un compuesto de la Fórmula I, que incluye en una realización especifica de la presente divulgación, el compuesto de la Fórmula I es compuesto 1 o la sal de malato del compuesto 1 a un paciente, quien ha sido identificado o diagnosticado como el que tiene enfermedad relacionada con fusión en ROS1, tales como, NSCLC positivo en la fusión de SLC34A2-ROS1, NSCLC positivo en la fusión de CD74-ROS1 y NSCLC positivo en la fusión de FIG-ROS1.
[0123] En otro aspecto de la presente divulgación, un compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo se administra a un paciente para prevenir o tratar un cáncer de pulmón. En algunas de estas realizaciones de la presente divulgación, el procedimiento comprende administrar al paciente, que lo necesita, una composición que comprende al menos un inhibidor de proteína de fusión ROS1, al menos un inhibidor del gen de fusión ROS1 que codifica la proteína de fusión, al menos un inhibidor de un gen que codifica ROS1 o una combinación de los mismos, como un ingrediente activo.
ROS1 y Socios de Fusión en SLC34A2, CD74 y FIG
[0124] La proteína ROS1, la proteína SLC34A2, la proteína CD74 y la proteína FIG, la fusión en SLC34A2-ROS1 o la fusión en CD74- ROS1; o la fusión en FIG-ROS1 o a veces llamada simplemente la "proteína de fusión ROS1 y ácidos nucleicos," que incluyen procedimientos para detectar, diagnosticar, equipos para detectar y diagnosticar, procedimientos de cribado, procedimientos de tratamiento y prevención y varias terapias para pacientes con cáncer de pulmón, procedimientos para medir la eficacia de tratamientos y otros ingredientes farmacéuticos que se pueden utilizar en combinación con varios tratamientos descritos a continuación.
[0125] La proteína ROS1 es un receptor transmembrana huérfano de tirosina quinasa. La proteína quinasa ROS1 humana (codificada por el gen ROS1) que es un receptor de tirosina quinasa largo de 2347 aminoácidos que es propenso a expresión aberrante que provoca cáncer. Una descripción de quinasa ROS1 humana de longitud completa (con la secuencia de aminoácidos de la proteína ROS1 humana) se puede encontrar en UniProt Número de Adhesión P08922(E.C.- 2.7.10.1). Tal como se muestra en la Tabla 1, el péptido señal, extracelular, transmembrana y dominios quinasa de ROS1 se encuentran en los siguientes residuos de aminoácidos en SEQ ID NO: 1
Tabla 1. Residuos de aminoácidos y dominios de proteína ROS1 en SEQ ID NO: 1
[0126] La proteína ROS1 humana se puede codificar utilizando un gen ROS1 situado en cromosoma 6 humano. El dominio C-terminal de proteína ROS1 puede incluir una secuencia de aminoácidos codificada por un polinucleótido desde el 31° o 32° o 34° exón hasta el último exón (por ejemplo, 32° o 34° exón) o un fragmento del mismo de gen ROS1. El dominio C-terminal de proteína ROS1 puede incluir de manera consecutiva al menos aproximadamente 100 aminoácidos desde la posición inicial del 31° o 32° exón. Por ejemplo, el dominio C-terminal de proteína ROS1 puede incluir de manera consecutiva aproximadamente 100 hasta aproximadamente 700 aminoácidos, de manera consecutiva aproximadamente 200 hasta aproximadamente 600 aminoácidos o de manera consecutiva aproximadamente 250 hasta aproximadamente 500 aminoácidos, o de manera consecutiva aproximadamente 270 hasta aproximadamente 425 aminoácidos, o de manera consecutiva aproximadamente 270 hasta aproximadamente 450 aminoácidos, o de manera consecutiva aproximadamente 475 hasta aproximadamente 625 aminoácidos desde la posición inicial del 32° exón (forma larga) o el 34° exón (forma corta) hacía C-terminal de la proteína ROS1. El punto de fractura para la proteína ROS1 es 1750 o 1852-1853 aa para proteínas de fusión SLC34A2- ROS1 y proteínas de fusión CD74-ROS1 y 1880-1881 aa para proteínas de fusión FIG-ROS1.
[0127] En una realización de ejemplo de la presente divulgación, el gen SLC34A2 humano que codifica la proteína SLC34A2 humana (ARNm NM_001177998) se encuentra en cromosoma 4 humano (4p15.2) y tiene 13 exones y una longitud de aminoácidos de aproximadamente 690 aminoácidos. La proteína que se codifica por este gen es un transportador de fosfato dependiente de sodio sensible a pH. Los defectos en este gen son una causa de microlitiasis alveolar pulmonar. Para este gen se descubrieron tres variantes de transcripción que codifican dos isoformas diferentes. La proteína SLC34A2 se puede extraer de un mamífero, tal como un humano. El dominio N-terminal de una proteína de fusión SLC34A2-ROS1 puede incluir una secuencia de aminoácidos, que se codifica por un polinucleótido del primer exón hasta el 12° exón o desde el primer exón hasta el cuarto exón, o desde el primer exón hasta el segundo exón del gen SLC34A2. Estos últimos exones observados en la estructura del gen SLC34A2 tienen puntos de rotura inferidos de 429 y 2076. El domino N-terminal de una proteína de fusión SLC34A2-ROS1 puede incluir de manera consecutiva al menos aproximadamente 30-250 aminoácidos desde la 1a posición (que es al menos una secuencia de aminoácidos desde la 1a hasta 250a posición o fragmentos de la misma) de la proteína SLC34A2. El dominio N-terminal de una proteína de fusión SLC34A2-ROS1 puede incluir de manera consecutiva aproximadamente 30 hasta 225 aminoácidos, de manera consecutiva aproximadamente 40 hasta 200 aminoácidos o de manera consecutiva aproximadamente 40 hasta 175 aminoácidos desde la 1a posición de aminoácido de la proteína SLC34A2.
[0128] En una realización de ejemplo de la presente divulgación, la fusión SLC34A2-ROS1 es una translocación cromosómica entre cromosomas 6 y 4 humanos que une la región 3' de ROS1 a la región 5' de SLC34A2. La proteína de fusión se detecta y se valida utilizando varios procedimientos conocidos por un experto en la técnica o como se describe en el presente documento. En algunas realizaciones de la presente divulgación, un compuesto de la Fórmula I o una sal farmacéuticamente aceptable del mismo se administra a continuación a un paciente que lo necesita para prevenir o tratar un cáncer de pulmón. En algunas realizaciones de la presente divulgación, una composición se administra a un paciente, que la necesita, la composición comprende al menos un inhibidor de proteína de fusión SLC34A2-ROS1, al menos un inhibidor del gen de fusión SLC34A2-ROS1 que codifica la proteína de fusión, al menos un inhibidor de un gen que codifica ROS1 o una combinación de los mismos, como un ingrediente activo.
[0129] En la proteína de fusión SLC34A2-ROS1, la fusión o la región de fusión puede aparecer entre varios exones del gen SLC34A2 y el gen ROS1. Un experto en la técnica conoce muchas fusiones. Los ejemplos de tales fusiones incluyen el 2° y/o el 4° exón del gen SLC34A2 y el 32° (Largo, L) y/o el 34° exón (Corto, S) del gen ROS1, al que se hace referencia como un punto de fusión o un punto de rotura. Se conocen otras fusiones o puntos de rotura en ROS1 y SLC34A2, y se pueden encontrar en el Catálogo COSMIC de mutaciones somáticas en base de datos de cáncer v.68 en cancer.sanger.ac.uk/cancergenome/projects/cosmic/. El término “una región de fusión” puede hacer referencia a un fragmento de polinucleótido (aproximadamente 10-30 nucleótidos) o un fragmento de polipéptido (aproximadamente 5-30 aminoácidos) alrededor del punto de fusión.
[0130] La fusión CD74-ROS1 es una translocación cromosómica que involucra ROS1 entre cromosomas 5 y 6 humanos que une la región 3' de ROS1 a la región 5' de CD74. La proteína de fusión se detecta y se valida utilizando
varios procedimientos conocidos por un experto en la técnica o como se describe en el presente documento. El dominio N-terminal de una proteína de fusión CD74-ROS1 puede incluir una secuencia de aminoácidos que se codifica por un polinucleótido del primer exón hasta el 12° exón o desde el primer exón hasta el cuarto exón, o desde el primer exón hasta el segundo exón del gen CD74. El domino N-terminal de una proteína de fusión CD74-ROS1 puede incluir de manera consecutiva al menos aproximadamente 30-250 aminoácidos desde la 1a posición (que es al menos una secuencia de aminoácidos desde la ia hasta 250a posición o fragmentos de la misma) de la proteína CD74. El dominio N-terminal de una proteína de fusión CD74-ROS1 puede incluir de manera consecutiva aproximadamente 30 hasta 225 aminoácidos, de manera consecutiva aproximadamente 40 hasta 200 aminoácidos o de manera consecutiva aproximadamente 40 hasta 210 aminoácidos o fragmentos de los mismos desde la ia posición de aminoácido de la proteína CD74.
[0131] En la proteína de fusión, la fusión o la región de fusión puede aparecer entre varios exones del gen CD74 y el gen ROS1. Un experto en la técnica conoce muchas fusiones. Los ejemplos de tales fusiones en CD74-ROS1 incluyen el 6° exón del gen CD74 unido al 32° o 34° exón del gen ROS1, al que se hace referencia como un punto de fusión o un punto de rotura. Se conocen otras fusiones o puntos de rotura en ROS1 y CD74, y se pueden encontrar en el Catálogo COSMIC de mutaciones somáticas en base de datos de cáncer v.68 en cancer.sanger.ac.uk/cancergenome/projects/cosmic/. El término “una región de fusión” puede hacer referencia a un fragmento de polinucleótido (aproximadamente 10-30 nucleótidos) o un fragmento de polipéptido (aproximadamente 5-30 aminoácidos) alrededor del punto de fusión.
[0132] La proteína de fusión FIG-ROS1 es el resultado de una eliminación intracromosómica en cromosoma 6 humano con fusión de la región 5' de FIG (aka GOPC) con la región 3' de ROS1. Las fusiones FIG-ROS1 se han identificado en muestras de pacientes con colangiocarcinoma y cáncer ovárico con una frecuencia de 8,7% y 0,5% respectivamente. La proteína de fusión FIG-ROS1 se puede detectar y validar utilizando varios procedimientos conocidos por un experto en la técnica o como se describe en el presente documento. El domino N-terminal de una proteína de fusión FIG-ROS1 puede incluir de manera consecutiva al menos aproximadamente 150-500 aminoácidos desde la 1a posición (que es al menos una secuencia de aminoácidos desde la 1a hasta 250a posición o fragmentos de la misma) de la proteína FIG. El dominio N-terminal de proteína de fusión FIG-ROS1 puede incluir de manera consecutiva aproximadamente 150 hasta aproximadamente 450 aminoácidos, de manera consecutiva aproximadamente 220 hasta 425 aminoácidos o de manera consecutiva aproximadamente 220 hasta aproximadamente 420 aminoácidos o fragmentos de los mismos desde la 1a posición de aminoácido de la proteína FIG.
[0133] En la proteína de fusión FIG-ROS1, la fusión o la región de fusión puede aparecer entre varios exones del gen FIG y el gen ROS1. Un experto en la técnica conoce varias fusiones FIG-ROS1. Los ejemplos de tales fusiones incluyen el 4° o el 8° exón del gen FIG unido al 35° o 36° exón del gen ROS1, al que se hace referencia como un punto de fusión o un punto de rotura. Se conocen otras fusiones o puntos de rotura en ROS 1 y FIG, y se pueden encontrar en el Catálogo COSMIC de mutaciones somáticas en base de datos de cáncer v.68 en cancer.sanger.ac.uk/cancergenome/projects/cosmic/. El término “una región de fusión” puede hacer referencia a un fragmento de polinucleótido (aproximadamente 10-30 nucleótidos) o un fragmento de polipéptido (aproximadamente 5-30 aminoácidos) alrededor del punto de fusión.
[0134] Otros socios de fusión ROS 1 pueden incluir TPM3, SDC4, EZR y LRIG3, además de las fusiones SLC34A2, CD74 y FIG descritas detalladamente en el presente documento. Ver la Figura 1 en el presente documento, por ejemplo, Takeuchi K, Soda M, Togashi Y, Suzuki R, Sakata S, Hatano S, et al. RET, ROS1 and ALK fusions in lung cancer. Nat Med. 2012, y Kurtis D. Davies, Anh T. Le, Mariana F. Theodoro, Margaret C. Skokan, Dara L. Aisner, Eamon M. Berge, Luigi M. Terracciano, Matteo Incarbone, Massimo Roncalli, Federico Cappuzzo, D. Ross Camidge, Marileila Varella-Garcia y Robert C. Doebelet, Identifying and Targeting ROS1 Gene Fusions in Non-Small Cell Lung Cancer, (2012), Clin Cancer Res. 1 de septiembre de 2012; 18(17): 4570-4579.
[0135] En una realización, la proteína de fusión ROS1 de la presente divulgación comprende el dominio C-terminal de proteína ROS1 humano (por ejemplo, ROS1 humano tal como se proporciona en SEQ ID NO.1), que proporciona un dominio quinasa funcional, que consiste esencialmente en aproximadamente 50 hasta 300 aminoácidos consecutivos empezando desde un aminoácido, que corresponde a la posición inicial del 32° exón de la proteína ROS1 y, a continuación, hacía C-terminal de la proteína ROS1.
[0136] Como se usa en el presente documento, el número de exón se cuenta según el número de exón adjudicado por el Centro Nacional para la Información Biotecnológica (NCBI) albergado por los Institutos Nacionales de Salud, Bethesda, Maryland, EEUU: En algunas realizaciones de ejemplo, la proteína de fusión SLC34A2-ROS1 puede tener cualquiera de las secuencias de aminoácidos identificadas como tal por el NCBI. Las secuencias de nucleótidos de moléculas de ADN y las secuencias de aminoácidos de proteínas codificadas por las moléculas de ADN se pueden determinar utilizado un secuenciador de ADN automático o un secuenciador de péptido automático. Las secuencias (de nucleótidos o aminoácidos) determinadas por tales medios de secuenciación automática pueden incluir error parcial en comparación con secuencias reales. Generalmente las secuencias determinadas por secuenciación automática pueden tener identidad de secuencia de al menos aproximadamente 90%, de al menos aproximadamente 95%, de al menos aproximadamente 99%, de al menos aproximadamente 99,9% en comparación con secuencias
reales. Por lo tanto, la proteína de fusión, el gen de fusión o la región de fusión puede tener una secuencia de aminoácidos o una secuencia de nucleótidos, que tiene una identidad de secuencia de al menos aproximadamente 90%, al menos aproximadamente 95%, al menos aproximadamente 99,9% en comparación con la secuencia de identificados como tales por el NCBI.
[0137] Con el fin de simplicidad, la siguiente divulgación se refiere a la proteína de fusión ROS1 en SLC34A2-ROS1, pero puede aplicarse a otras proteínas de fusión ROS1 descritas en el presente documento. La proteína de fusión SLC34A2-ROS1 representada, en algunas realizaciones de la divulgación puede consistir en 80-200 residuos de aminoácidos N-terminales de SLC34A2 y al menos 300 residuos C-terminales de ROS1, preferiblemente que oscilan entre 300-500 residuos de aminoácidos C-terminales. El gen de fusión tiene un dominio tirosina quinasa en conjunto con dominios helicoidales. Aunque no se desea ceñirse a la teoría, se cree que la conformación terciaria de la proteína de fusión SLC34A2-ROS1 induce homodimerización, que activará el dominio tirosina quinasa oncogénico mediante autofosforilación. En conjunto, el gen de fusión SLC34A2-ROS1 puede expresarse altamente y, a continuación, dimerizarse después de desplazamiento que se debe a SLC34A2. A continuación, el dominio tirosina quinasa de proteína ROS1 dimerizada se puede simular excepcionalmente, por lo tanto, facilitando la simulación de una vía oncogénica, por ejemplo, tal como se ve en una fusión ROS1 relacionada con NSCLC.
[0138] Una vez la fusión es detectada, se conocerá como un gen de fusión de SLC34A2-ROS1, que codifica la proteína de fusión de SLC34A2-ROS1. En el presente documento se da a conocer un procedimiento para proporcionar información para diagnosticar un cáncer de pulmón, que comprende la etapa de detección, en una muestra de prueba obtenida de un sujeto, de una fusión, como se describe en el presente documento. El diagnóstico compararía un gen de fusión, que codifica la proteína de fusión; y una sobreexpresión de ROS1 comparada con una muestra estándar de un individuo sin un cáncer, donde al menos un elemento es seleccionado y detectado en la muestra de prueba, que permite al sujeto ser identificado como cualquier o todos de los siguientes: un paciente de cáncer, un paciente con cáncer de pulmón, un paciente con NSCLC, un paciente con NSCLC relacionado con fusión ROS1 y/o un paciente con NSCLC relacionado con fusión SLC34A2-r Os 1.
[0139] Se puede detectar la eliminación, la inversión o la translocación de cromosoma 6 utilizando un polinucleótido (una sonda) capaz de hibridar con (complementariamente unido a) la deleción, la inversión o la translocación en cromosoma 6 humano y/o un primer par capaz de detectar la eliminación, la inversión o la translocación de cromosoma 6 humano, por ejemplo, capaz de producir un fragmento de polinucleótido que tiene desde 100 hasta 200 nucleótidos consecutivos, que incluyen la región de inversión en cromosoma 6 humano. La proteína de fusión, el gen de fusión y la región de fusión se describen en el presente documento. En una realización de ejemplo de la presente divulgación, se puede detectar también la proteína de fusión detectando la presencia de la proteína de fusión o el gen de fusión o ARNm correspondiente al gen de fusión, por ejemplo, con el uso de anticuerpos ROS1 específicos o de fusión ROS1 específicos conocidos en la técnica y descritos en el presente documento.
[0140] La presencia de la proteína de fusión se puede detectar utilizando un ensayo general, que mide la interacción entre la proteína de fusión y un material (por ejemplo, un anticuerpo o un aptámero) específicamente unido a la proteína de fusión. El ensayo general puede ser inmunocromatografía, tinción inmunohistoquímica, ensayo por inmunoabsorción ligado a enzimas (ELISA), radioinmunoensayo (RIA), inmunoensayo enzimático (EIA), inmunoensayo fluorescente (FIA), inmunoensayo luminiscente (LIA), transferencia western, FACS y los similares.
[0141] Adicionalmente, la presencia del gen de fusión o el ARNm se puede detectar utilizando un ensayo general, tal como PCR, FISH (hibridación in situ fluorescente) y los similares, utilizando un polinucleótido capaz de hibridar con (complementariamente unido a) el gen de fusión o el ARNm. FISH se describe más detalladamente a continuación. Se puede detectar y/o validar el gen de fusión utilizando las técnicas de integración de secuenciación de transcriptoma entero (ARN) y/o ADN de genoma entero por medio de tecnologías de secuenciación masiva en paralelo. El polinucleótido capad de hibridar con el gen de fusión o el ARNm puede ser un siARN, un oligonucleótido, una sonda de ADN o cebador de ADN, que puede detectar el gen de fusión o el ARNm utilizando una hibridación directa con el gen o la transcripción fusionado o truncado en la muestra de prueba.
[0142] En otras realizaciones, la hibridación in situ fluorescente (FISH) se emplea en los procedimientos de la presente divulgación (como se describe en Verma et al. "Human Chromosomes: A Manual Of Basic Techniques", Pergamon Press, New York, N.Y. (1988)). Un ensayo FISH se puede llevar a cabo utilizando uno o más conjunto de sondas, las realizaciones se proporcionan tal como: (1) Un primer conjunto de sondas, que es un primer conjunto de sondas dirigido a sitio cromosómico que contiene el gen ROS1 (primer sitio cromosómico); consiste en una sonda 1A etiquetada con una primera sustancia fluorescente y una sonda 1B etiquetada con una segunda sustancia fluorescente; la sonda 1A es complementaria para la primera región, que es la región 5' en el primer sitio cromosómico mencionado anteriormente, la sonda 1B es complementaria para la segunda región, que está a una distancia de la primera región mencionada anteriormente y es la región 3' en el primer sitio cromosómico mencionado anteriormente y el punto de rotura en el gen ROS1, cuando el gen de fusión SLC34A2-ROS1 se produce por una translocación entre los genes SLC34A2 y ROS1 está ubicado en la cola 3' de la primera región mencionada anteriormente, entre la primera y la segunda regiones mencionadas anteriormente o en la cola 5' de la segunda región mencionada anteriormente; (2) Un segundo conjunto de sondas, que es un segundo conjunto de sondas dirigido a sitio cromosómico que contiene el gen SLC34A2 (segundo sitio cromosómico); consiste en una sonda 2A etiquetada con una primera sustancia
fluorescente y una sonda 2B etiquetada con una segunda sustancia fluorescente; la sonda 2A es complementaria para la primera región, que es la región 5' en el segundo sitio cromosómico mencionado anteriormente, la sonda 2B es complementaria para la segunda región, que está a una distancia de la primera región mencionada anteriormente y es la región 3' en el segundo sitio cromosómico mencionado anteriormente y el punto de rotura en el gen SLC34A2, cuando el gen de fusión SLC34A2-ROS1 se produce por una translocación entre los genes SLC34A2 y ROS1 está ubicado en la cola 3' de la primera región mencionada anteriormente, entre la primera y la segunda regiones mencionadas anteriormente o en la cola 5' de la segunda región mencionada anteriormente; (3) un tercer conjunto de sondas que consiste en la sonda 2A mencionada anteriormente y la sonda 1B mencionada anteriormente; y (4) un cuarto conjunto de sondas que consiste en una sonda 4A, que es complementaria para el sitio cromosómico, que contiene el gen ROS1 (el tercer sitio cromosómico) y una sonda 4B, que es complementaria para el sitio cromosómico, que contiene el gen SLC34A2 (el cuarto sitio cromosómico).
[0143] La longitud del primer sitio cromosómico mencionado anteriormente puede ser de 0,5-2,0 Mb. La longitud del segundo sitio cromosómico mencionado anteriormente puede ser de 0,5-2,0 Mb. La longitud del tercer sitio cromosómico mencionado anteriormente puede ser de 0,5-2,0 Mb. La longitud del cuarto sitio cromosómico mencionado anteriormente puede ser de 0,5-2,0 Mb.
[0144] En algunas realizaciones, el ensayo FISH puede relacionarse con otras técnicas de mapeo cromosómico físico y datos de mapa genético. La técnica FISH se conoce bien (véanse, por ejemplo, Patentes de Estados Unidos Números 5.756.696; 5.447.841; 5.776.688; y 5.663.319). Se pueden encontrar los ejemplos de datas de mapa genético en la Publicación Genómica de Ciencia del 1994 (265: 1981f). La relación entre la localización del gen, que codifica proteína ROS1 y/o, en el caso de polipéptidos de fusión ROS1, el gen, que codifica al socio de fusión de una proteína de fusión ROS1 (por ejemplo, el gen FIG, el gen SLC34A2 o el gen CD74) en un mapa cromosómico físico y una enfermedad específica o una predisposición a una enfermedad específica puede ayudar a delimitar la región del ADN asociado con aquella enfermedad genética. Las secuencias de nucleótidos de la presente divulgación se pueden usar para detectar diferencias en secuencias de genes entre individuos normales, portadores o afectados.
[0145] La hibridación in situ de preparaciones cromosómicas y técnicas de mapeo físico, tales como, análisis de enlaces, que utiliza marcadores cromosómicos establecidos, se puede utilizar para mapas genéticos extendidos. A menudo la disposición de un gen en el cromosoma de otra especie de mamíferos, tal como, de ratón, puede revelar marcadores asociados, aunque el número o la sección de un cromosoma humano particular se desconoce. Se puede asignar nuevas secuencias a secciones cromosómicas o partes de las mismas utilizando mapeo físico. Esto proporciona información valiosa a investigadores, que buscan genes de enfermedades utilizando clonación posicional u otras técnicas de descubrimiento genético. Cuando la enfermedad o el síndrome se ha localizado de manera aproximada por enlace genético a una región genómica particular, por ejemplo, AT con 1q22-23 (Gatti et al., Nature 336: 577-580 (1988)), cualquier mapeo de secuencias en aquel área puede representar genes asociados o reguladores para una investigación adicional. Las secuencias de nucleótidos que codifican las fusiones ROS 1 de la presente divulgación o porciones de las mismas se pueden utilizar también para detectar diferencias en la localización cromosómica debida a translocación, inversión, etc., entre individuos normales, portadores o afectados.
[0146] Se debe entender que todos los procedimientos (por ejemplo, PCR y FISH), que detectan los polinucleótidos que codifican un polipéptido con actividad quinasa ROS1 (por ejemplo, polinucleótidos con ROS1 de longitud completa o con fusión ROS1, tales como, SLC34A2- ROS1, o CD74-ROS1, o FIG-ROS1(S)), se pueden combinar con otros procedimientos para detectar polinucleótidos con actividad quinasa ROS1 o polinucleótidos que codifican un polipéptido con actividad quinasa ROS1. Por ejemplo, la detección de un polinucleótido de fusión FIG-ROS1 en el material genético de una muestra biológica (por ejemplo, en una célula tumoral circulante) se puede seguir utilizando el análisis de transferencia western o el análisis de inmunohistoquímica (IHC) de las proteínas de la muestra para determinar si el polinucleótido FIG-ROS1 se expresó realmente como un polipéptido de fusión FIG-ROS1 en la muestra biológica. Tales análisis de transferencia western o IHC se pueden llevar a cabo utilizando un anticuerpo que se une de forma específica al polipéptido codificado por el polinucleótido FIG-ROS1 detectado, o el análisis se puede llevar a cabo utilizando anticuerpos que se unen de manera específica o a FIG de longitud completa (por ejemplo, se unen al N-terminal de la proteína) o como a ROS1 de longitud completa (por ejemplo, unen un epítopo en el dominio quinasa de ROS1). Tales técnicas se conocen en la técnica (ver, por ejemplo, la Patente de EEUU Número 7.468.252).
[0147] En otro ejemplo, la tecnología CISH de Dako permite hibridación in situ fluorescente con inmunohistoquímica en la misma sección de tejido. Véanse, Elliot et al., Br J Biomed Sci 2008; 65(4): 167-171,2008 para una comparación de CISH y FISH.
[0148] Otro aspecto de la presente divulgación da a conocer un procedimiento para diagnosticar a un paciente que tiene un cáncer de pulmón o un posible cáncer de pulmón provocado por una quinasa ROS1. El procedimiento incluye ponerse en contacto con una muestra biológica de dicho cáncer de pulmón o un posible cáncer de pulmón (donde la muestra biológica comprende al menos una molécula de ácido nucleico) utilizando una sonda que hibrida bajo condiciones estrictas con una molécula de ácido nucleico que codifica un polipéptido con actividad quinasa ROS1, tales como, un polipéptido ROS1 de longitud completa o un polipéptido con fusión ROS1 (por ejemplo, un polinucleótido con fusión FIG-ROS1, un polinucleótido con fusión SLC34A2-ROS1 o un polinucleótido con fusión CD74-ROS1) y donde la hibridación de dicha sonda con al menos una molécula de ácido nucleico en dicha muestra
biológica identifica a dicho paciente como el que tiene un cáncer de pulmón o un posible cáncer de pulmón provocado por una quinasa ROS1.
[0149] Otro aspecto más de la presente divulgación da a conocer un procedimiento para diagnosticar a un paciente que tiene un cáncer de pulmón o un posible cáncer de pulmón provocado por una quinasa ROS 1. El procedimiento incluye ponerse en contacto con una muestra biológica de dicho cáncer de pulmón o un posible cáncer de pulmón (donde dicha muestra biológica comprende al menos un polipéptido) utilizando un reactivo que se une de manera específica con un polipéptido con actividad quinasa ROS1 (por ejemplo, un polipéptido con fusión FIG-ROS1, un polipéptido con fusión SLC34A2-ROS1 o CD74-ROS1), donde la unión específica de dicho reactivo con al menos un polipéptido de fusión en dicha muestra biológica identifica a dicho paciente como el que tiene un cáncer de pulmón o un posible cáncer de pulmón provocado por una quinasa ROS1.
[0150] En varias realizaciones de la presente divulgación, la identificación de un cáncer de pulmón o un posible cáncer de pulmón, como el que se provoca por una quinasa ROS1, determinará al paciente, que tiene cáncer de pulmón o un posible cáncer de pulmón, como probable que responda a un terapéutico que inhibe ROS1.
[0151] Un equipo para detectar translocaciones entre los genes SLC34A2 y ROS1 puede incluir uno o más conjunto de sondas. (1) Un primer conjunto de sondas incluye una sonda 1A etiquetada con una primera sustancia fluorescente y una sonda 1B etiquetada con una segunda sustancia fluorescente; la sonda 1A es complementaria para la primera región, que es la región 5' en el primer sitio cromosómico mencionado anteriormente, la sonda 1B es complementaria para la segunda región, que está a una distancia de la primera región mencionada anteriormente y es la región 3' en el primer sitio cromosómico mencionado anteriormente y el punto de rotura en el gen ROS 1, cuando el gen de fusión SLC34A2-ROS1 se produce por una translocación entre los genes SLC32A2 y ROS 1, está ubicado en la cola 3' de la primera región mencionada anteriormente, entre la primera y la segunda regiones mencionadas anteriormente o en la cola 5' de la segunda región mencionada anteriormente; (2) Un segundo conjunto de sondas, que es un segundo conjunto de sondas dirigido a sitio cromosómico que contiene el gen SLC32A2 (segundo sitio cromosómico); consiste en una sonda 2A etiquetada con una primera sustancia fluorescente y una sonda 2B etiquetada con una segunda sustancia fluorescente; la sonda 2A es complementaria para la primera región, que es la región 5' en el segundo sitio cromosómico mencionado anteriormente, la sonda 2B es complementaria para la segunda región, que está a una distancia de la primera región mencionada anteriormente y es la región 3' en el segundo sitio cromosómico mencionado anteriormente y el punto de rotura en el gen SLC32A2, cuando el gen de fusión SLC34A2-ROS1 se produce por una translocación entre los genes SLC32A2 y ROS1, está ubicado en la cola 3' de la primera región mencionada anteriormente, entre la primera y la segunda regiones mencionadas anteriormente o en la cola 5' de la segunda región mencionada anteriormente; (3) Un tercer conjunto de sondas que consiste en la sonda 2A mencionada anteriormente y la sonda 1B mencionada anteriormente; y un cuarto conjunto de sondas que consiste en una sonda 4A que es complementaria para el sitio cromosómico, que contiene el gen ROS1 (el tercer sitio cromosómico) y una sonda 4B, que es complementaria para el sitio cromosómico, que contiene el gen SLC32A2 (el cuarto sitio cromosómico).
[0152] Un equipo útil para identificar pacientes susceptibles a translocación SLC34A2-ROS1 incluye uno o más elementos seleccionados de un grupo que comprende una explicación del uso de las sondas, un colorante de contraste de ADN, un tampón para hibridación, un encapsulante y una lámina de control. El equipo hace posible implementar de manera conveniente y eficiente el procedimiento de detección de la presente divulgación. El equipo puede incluir como elementos necesarios (ingredientes esenciales) el primer conjunto de sondas mencionado anteriormente, el segundo conjunto de sondas, el tercer conjunto de sonda o el cuarto conjunto de sondas. Se pueden incluir dos o más tipos de conjuntos de sondas en el equipo. Por ejemplo, el equipo puede incorporar el primer conjunto de sondas y el tercer conjunto de sondas. Dado que los detalles para cada conjunto de sondas se han descrito anteriormente, no se repetirán aquí.
[0153] La detección de la presencia o la ausencia de un polinucleótido de fusión SLC34A2-ROS1 se puede llevar a cabo directamente utilizando ADN genómico que codifica el polipéptido de fusión mencionado anteriormente o una transcripción de aquel ADN genómico, pero también se puede llevar a cabo de forma indirecta utilizando un producto de traducción de aquella transcripción (el polipéptido de fusión mencionado anteriormente).
[0154] Puesto que el ADN genómico que codifica el polipéptido de fusión se forma mediante inversión entre la región 117.61-117.75Mb de cromosoma 6, (6q22) se puede detectar el fenómeno de la presente divulgación en la “detección de la presencia o la ausencia de un polinucleótido de fusión SLC34A2-ROS1”. En esta detección de inversión, por ejemplo, se puede detectar una rotura entre una región del lado 5' arriba de la región codificante de dominio quinasa del gen ROS1 y una región del lado 3' abajo de aquella región codificante del gen ROS1, o se puede detectar una rotura entre la región codificante de una parte o todos los dominios helicoidales y región de lado 5' arriba de aquella región codificante del gen SLC32A2 y una región del lado 3' abajo de la región codificante de los dominios helicoidales del gen SLC32A2.
[0155] Se pueden utilizar las técnicas conocidas y disponibles para un experto en la "detección de la presencia o la ausencia de un polinucleótido de fusión SLC34A2-ROS1" en la presente divulgación. Si “ADN genómico que codifica el polipéptido de fusión mencionado anteriormente” es el objeto, se puede utilizar, por ejemplo, hibridación in situ (ISH) que utiliza fluorescencia o similares, PCR genómico, secuenciación directa, mancha southern o análisis de microarray
genómico. Si una “transcripción del ADN genómico mencionado anteriormente” es el objeto, se puede utilizar, por ejemplo, RT-PCR, secuenciación directa, mancha northern, dot blot o análisis de microarray de ADNc.
[0156] El equipo de la presente divulgación puede incluir también otros elementos. Los ejemplos de estos otros elementos son la especificación para el uso de las sondas, un colorante de contraste secundario de ADN, tal como, DAPI, un tampón de hibridación, un tampón de lavado, disolventes, medio de fijación, láminas de control, frascos de reacción y otro equipamiento. Se pueden incluir también las especificaciones con fines diagnósticos. Además, se pueden incluir las especificaciones, etc., que muestran como organizar la detección (identificación positiva) de translocación SLC34A2-ROS1 en muestras cromosómicas de pacientes con cáncer para estos pacientes utilizando un inhibidor de quinasa ROS1. Además, se pueden incluir un plan para determinar el curso de tratamiento y una explicación de este plan.
[0157] Se contemplan sondas comparativamente largas (aproximadamente 200 kb (sonda 1A) hasta aproximadamente 1,370 kb (sonda 4B). Por lo tanto, la complementación entre las sondas y las secuencias diana no tiene que ser altamente restrictiva, en la medida en que se logra la hibridación específica deseada en esta divulgación. Un ejemplo de similaridad entre secuencias diana es al menos 90%, preferiblemente al menos 95% y más preferiblemente al menos 98%.
[0158] En el caso cuando se usan un primer conjunto de sondas y un segundo conjunto de sondas, las dos fluorescentes se separan y se detectan de manera individual en muestras cromosómicas, en las que se produce la translocación entre el gen SLC34A2 y el gen ROS1; en muestras cromosómicas en las que la translocación no se produce, se observan típicamente las dos señales fluorescentes una al lado de la otra o se observa una señal (amarilla), que es una combinación de las dos señales fluorescentes. Por lo tanto, la presencia o la ausencia de una translocación entre el gen SLC34A2 y el gen ROS1 se refleja en el patrón de la señal fluorescente. Entonces, se puede determinar una translocación entre el gen SLC34A2 y el gen ROS1 a partir del patrón de la señal fluorescente.
[0159] La opinión alcanzada anteriormente se basa preferiblemente y típicamente en una comparación de un resultado con un control (muestra de prueba). Aquí, el control es una muestra cromosómica derivada de un paciente con cáncer de pulmón de células no pequeñas o una muestra cromosómica derivada de un paciente, que muestra lesiones precancerosas. Adicionalmente, se pueden utilizar las muestras cromosómicas de un paciente sin lesiones precancerosas, las muestras cromosómicas de pacientes, que no tienen cáncer, o las muestras cromosómicas recogidas de sujetos normales, sanos como el control. También se pueden utilizar las muestras cromosómicas derivadas de cepas celulares como un control.
[0160] Cuando el gen de fusión es un gen de fusión SLC34A2-ROS1, que codifica la proteína de fusión SLC34A2-ROS1, se puede detectar el gen de fusión SLC34A2-ROS1 (o cualquier otro gen de fusión ROS1, por ejemplo, CD74-ROS1, & FIG-ROS1) utilizando un polinucleótido (una sonda) capaz de hibridar con (unirse complementariamente con) la región de fusión y/o un primer par capaz de producir un fragmento de polinucleótido, que tiene de 100 hasta 200 nucleótidos consecutivos incluyendo la región de fusión. Adicionalmente, en una realización de ejemplo, se puede detectar la proteína de fusión SLC34A2-ROS1 utilizando un anticuerpo o un aptámero, que se une de forma específica con la región de fusión de la proteína de fusión SLC34A2-ROS1.
[0161] Adicionalmente, se puede llevar a cabo la detección utilizando un ensayo de fusión, que es una combinación del procedimiento de hibridación in situ cromogénica (CISH) y el procedimiento de hibridación in situ con plata (SISH). El “punto de fusión” en la presente especificación se refiere a un punto, donde una porción derivada de los genes respectivos de SLC34A2, se fusiona con una porción derivada del gen ROS1.
[0162] El término "capaz de hibridar con la región de fusión (o la región de inversión)" puede hacer referencia a tener una secuencia complementaria o una secuencia que tiene identidad de secuencia de al menos 90% con la de la región de fusión (o la región de inversión). Otra realización da a conocer una combinación para diagnosticar un cáncer, que incluye uno o más seleccionados del grupo, que consiste en un polinucleótido capaz de hibridar con la región de fusión, un primer par capaz de producir un fragmento de polinucleótido, que tiene de 100 a 200 nucleótidos consecutivos incluyendo la región de fusión. También un polinucleótido capaz de hibridar con la región de inversión en cromosoma 6 humano, un primer par capaz de producir un fragmento de polinucleótido que tiene de 100 a 200 nucleótidos consecutivos incluyendo la región de inversión de cromosoma 6 humano y un anticuerpo o un aptámero que se une a la región de fusión. Otra realización proporciona un procedimiento de la proteína de fusión y/o el gen de fusión para diagnosticar cáncer. El paciente puede ser cualquier mamífero, por ejemplo, un primate, tal como, un humano o un mono, un roedor, tal como, un ratón o una rata y, en particular, un humano. Las muestras de prueba, adecuadas para el uso en cualquier ensayo mencionado, pueden ser una célula (por ejemplo, una célula de pulmón); un tejido (por ejemplo, un tejido pulmonar); fluido corporal (por ejemplo, sangre); ADN tumoral circulante, células tumorales circulantes. Las muestras se pueden recoger de cualquier manera conocida por un experto en la técnica, incluyendo recogida de la biopsia quirúrgica de tumor, una biopsia central de tumor, una succión con aguja fina de tumor, efusión pleural y otros procedimientos conocidos de separación de células y tejidos de pacientes. Por ejemplo, se puede llevar a cabo un ensayo FISH (descrito en el presente documento) en células tumorales circulantes.
[0163] El paciente puede estar recibiendo tratamientos o está planeando ser tratado con un inhibidor de quinasa. La
muestra de prueba puede incluir una célula derivada de una célula cancerígena human o un extracto de la misma.
[0164] Otra realización de la presente divulgación da a conocer un gen de fusión, que codifica la proteína de fusión, donde un gen codifica el dominio N-terminal de las posiciones de socio de fusión en extremo 5' y un gen codifica el dominio quinasa C-terminal de las posiciones de proteína ROS1 en extremo 3'. En una realización de ejemplo de la presente divulgación, cuando la proteína de fusión es la proteína SLC34A2-ROS1, el gen de fusión puede estar representado como gen SLC34A2-ROS1, donde un gen codifica el dominio N-terminal de las posiciones de SLC34A2 en extremo 5' y un gen codifica el dominio C-terminal de las posiciones de proteína ROS1 en extremo 3'.
[0165] Otra realización de la presente divulgación proporciona un vector de expresión, que incluye el gen de fusión y, opcionalmente, elementos de transcripción (por ejemplo, un promotor y los similares) unidos de manera operativa con el gen de fusión. Otra realización de la presente divulgación proporciona una célula transformante con el vector de expresión.
[0166] Los especímenes biológicos obtenidos en el curso de tratamiento o diagnóstico (muestras de biopsias, etc.) a menudo se fijan en formalina, pero en este caso, el uso de hibridación in situ es ventajoso debido a que el genoma de ADN, que es el objeto de detección, es estable en fijación con formalina y debido a que la sensibilidad de detección es alta.
[0167] En la hibridación in situ, se puede detectar el ADN genómico, que codifica un polipéptido de fusión SLC34A2-ROS1 en el espécimen biológico hibridando el polinucleótido de (a) o (b) enunciado a continuación, que tiene una longitud de cadena de al menos 15 bases de nucleótidos:
(a) un polinucleótido, que es al menos una sonda seleccionada del grupo que consiste en sondas, que hibridan con un polinucleótido, que codifica la proteína SLC34A2, y sondas, que hibridan con un polinucleótido que codifica la proteína ROS1
(b) un polinucleótido, que es una sonda que hibrida con un sitio de fusión de un polinucleótido, que codifica la proteína SLC34A2, y un polinucleótido, que codifica la proteína ROS1.
[0168] En la hibridación in situ, se puede detectar el ADN genómico, que codifica un polipéptido de fusión CD74-ROS1 en el espécimen biológico hibridando el polinucleótido de (a) o (b) enunciado a continuación, que tiene una longitud de cadena de al menos 15 bases:
(a) un polinucleótido, que es al menos una sonda seleccionada del grupo que consiste en sondas, que hibridan con un polinucleótido, que codifica la proteína CD74, y sondas, que hibridan con un polinucleótido, que codifica la proteína ROS1
(b) un polinucleótido, que es una sonda, que hibrida con un sitio de fusión de un polinucleótido, que codifica la proteína CD74, y un polinucleótido, que codifica la proteína ROS1.
[0169] En la hibridación in situ, se puede detectar el ADN genómico, que codifica un polipéptido de fusión FIG-ROS1 en el espécimen biológico hibridando el polinucleótido de (a) o (b) enunciado a continuación, que tiene una longitud de cadena de al menos 15 bases:
(a) un polinucleótido, que es al menos una sonda seleccionada del grupo que consiste en sondas, que hibridan con un polinucleótido, que codifica la proteína FIG, y sondas, que hibridan con un polinucleótido que codifica la proteína ROS1
(b) un polinucleótido, que es una sonda que hibrida con un sitio de fusión de un polinucleótido, que codifica la proteína FIG, y un polinucleótido, que codifica la proteína ROS1.
[0170] Sin embargo, la secuencia de ADN de un gen puede mutar en el medio natura (que no es artificial). Por consiguiente, tales variantes naturales pueden ser, también, el objeto de la presente divulgación (de manera similar de aquí en adelante).
[0171] El polinucleótido enunciado en (a) de la presente invención puede ser cualquier polinucleótido, siempre y cuando puede detectar la presencia del ADN genómico, que codifica la proteína de fusión ROS1 mencionada anteriormente en el espécimen biológico hibridando con un polinucleótido, que codifica el socio de unión ROS1, es decir, proteína SLC34A2, CD74 o FIG o un polinucleótido, que codifica la proteína ROS1, que son las secuencias de bases diana de aquel polinucleótido. Preferiblemente es un polinucleótido enunciado en (a1) hasta (a4) a continuación.
[0172] (a1) Una combinación de un polinucleótido, que hibrida con la región codificante de una parte o de todo el dominio helicoidal de exones 1-2 y región del lado 5 arriba de aquella región codificante del gen SLC34A2 (a la que también se refiere como "sonda 1 de SLC34A2 de 5'" de aquí en adelante) y un polinucleótido, que hibrida con la región codificante del dominio quinasa y la región del lado 3' abajo de aquella región codificante del gen ROS1 (a la que se refiere como "sonda 1 de ROS1 de 3'” de aquí en adelante).
[0173] (a2) Una combinación de un polinucleótido, que hibrida con la región del lado 5' arriba de la región codificante del dominio quinasa del gen ROS1 (a la que también se refiere como "sonda 1 de ROS1 de 5'" de aquí en adelante) y un polinucleótido, que hibrida con la región codificante del dominio quinasa y la región del lado 3' abajo de aquella región codificante del gen ROS1 (a la que se refiere como "sonda 1 de ROS1 de 3'” de aquí en adelante).
[0174] (a3) Una combinación de un polinucleótido que híbrida con la región codificante de la Fibronectina del tipo III 9 y la región del lado 5' arriba de aquella región del gen ROS1 (a la que también se refiere como "sonda 2 de ROS1 de 5'" de aquí en adelante) y un polinucleótido, que hibrida con la región codificadora del dominio transmembrana y la región del lado 3' abajo de aquella región codificadora del gen ROS1 (a la que se refiere como "sonda 2 de ROS1 de 3'” de aquí en adelante).
[0175] (a4) Una combinación de un polinucleótido, que hibrida con la región codificadora de una parte o de todo el dominio helicoidal y la región del lado 5' arriba de aquella región codificadora del gen SLC34A2 (a la que también se refiere como "sonda 1 de SLC34A2 de 5'" de aquí en adelante) y un polinucleótido, que hibrida con la región codificadora del dominio de bobina enrollada y la región del lado 3' abajo de aquella región codificadora del gen SLC34A2 (a la que se refiere como "sonda 1 de SLC34A2 de 3'” de aquí en adelante).
[0176] La región (secuencia de bases diana) con la que hibrida el polinucleótido de (a1) utilizado en hibridación in situ es, preferiblemente, una región a menos de 1.000.000 bases desde el sitio de fusión del gen SLC34A2 y el gen ROS1, debido a razones de especificidad de la secuencia de bases diana y la detección de sensibilidad, y la región con la que hibrida el polinucleótido de (a2) hasta (a4) utilizado en la hibridación in situ es, preferiblemente, una región a menos de 1.000.000 bases del punto de rotura en el gen SLC34A2 o el gen ROS1, por las mismas razones.
[0177] El polinucleótido enunciado en (a) o (b) anteriormente utilizado en la hibridación in situ es, preferiblemente, una recogida, que consiste en una pluralidad de tipos de polipéptido, que puede cubrir todas las secuencias de bases diana, por razones de especificidad de las secuencias de bases diana y la detección de sensibilidad. En este caso, la longitud de los polinucleótidos, que componen la recogida es de al menos 15 base y, preferiblemente, de 100 a 1000 bases.
[0178] El polinucleótido enunciado en (a) o (b) anteriormente utilizado en la hibridación in situ está etiquetado preferentemente con tinte fluorescente o similares para la detección. Los ejemplos de tal tinte fluorescente incluyen DEAC, FITC, R6G, TexRed y Cy5, pero no se limitan a ellos. Además de un tinte fluorescente, se puede etiquetar al polinucleótido mencionado anteriormente con un colorante (cromógeno), tal como, DAB o plata y los similares basándose en disposición metálica enzimática.
[0179] En hibridación in situ, si se usan la sonda 1 de SLC34A2 de 5' y la sonda 1 de ROS1 de 3', o si se usan la sonda 1 de ROS1 de 5' y la sonda 1 de ROS1 de 3', o si se usan la sonda 2 de ROS1 de 5' y la sonda 2 de ROS1 de 3', o si se usan la sonda 1 de SLC34A2 de 5' y la sonda 1 de SLC34A2 de 3', se prefiere que estas sondas se etiqueten con tintes diferentes una de la otra. Cuando se lleva a cabo la hibridación in situ utilizando una combinación de sondas etiquetadas con diferentes tintes de esta forma, cunado la señal producida por la etiqueta de la sonda 1 de SLC34A2 de 5' (por ejemplo, fluorescencia) y la señal se produce por la etiqueta de la sonda 1 de ROS1 de 3' en superposición, se puede determinar que se ha detectado el ADN genómico, que codifica un polipéptido de fusión SLC34A2-ROS1. Por otra parte, cuando la señal producida por la etiqueta de la sonda 1 de ROS1 de 5' y la señal producida por la etiqueta de la sonda 1 de ROS1 de 3' se dividen, o la señal producida por la etiqueta de la sonda 2 de ROS1 de 5' y la señal producida por la etiqueta de la sonda 2 de ROS1 de 3' se dividen, o la señal producida por la etiqueta de la sonda 1 de SLC34A2 de 5' y la señal producida por la etiqueta de la sonda 1 de SLC34A2 de 3' se dividen, se puede determinar que se ha detectado el ADN genómico, que codifica un polipéptido de fusión SLC34A2-ROS1.
[0180] El etiquetado de polinucleótidos se puede llevar a cabo utilizando una técnica conocida. Por ejemplo, se puede integrar una base de sustrato etiquetada con tinta fluorescente o los similares, utilizando una traducción de Nick o imprimación aleatoria, en un polinucleótido, etiquetando de este modo aquel polinucleótido. En la hibridación in situ, las condiciones utilizadas se pueden variar, cuando se hibridan el polinucleótido enunciado en (a) o (b) anteriormente y el espécimen biológico mencionado anteriormente, dependiendo de varios factores, tales como, la longitud del polinucleótido relevante, pero un ejemplo de condiciones de hibridación de alta exigencia es 0.2xSSC, 65°C, y un ejemplo de condiciones de hibridación de baja exigencia es 2.0xSSC, 50°C. Que conste, que las condiciones de hibridación de la misma exigencia como las condiciones mencionadas anteriormente se pueden alcanzar por un experto en la técnica seleccionando de manera apropiada las diferentes condiciones, tales como, concentración de sal (velocidad de disolución de SSC) y temperatura, así como concentración de tensioactivo (NP-40, etc.), concentración de formamida y pH. Por ejemplo, para las condiciones generales de hibridación para cribado, se proporcionan composiciones de polinucleótidos, que son capaces de hibridar en condiciones de moderada a alta exigencia con una secuencia de polinucleótido proporcionada en el presente documento o un fragmento de la misma o una secuencia complementaria de la misma. Las técnicas de hibridación se conocen bien en el ámbito de la biología molecular. Con fines ilustrativos, las condiciones adecuadas de exigencia moderada para poner a prueba la hibridación de un polinucleótido de la presente divulgación con otros polinucleótidos incluyen prelavado en una solución 5 veces SSC, 0,5% SDS, 1,0 mM Ed TA (pH 8.0); hibridar a 50°C-30°C, 5X SSC, durante la noche; seguido de lavado 2 veces a 65°C durante 20 minutos con cada uno de 2X, 0,5X y 0,2X SSC, que contiene 0,1% SDS. Un experto en la técnica entenderá que la exigencia de hibridación se puede manipular fácilmente, tal como, alterando el contenido de sal de la solución de hibridación y/o la temperatura con la que se lleva a cabo la hibridación. Por ejemplo, en otra realización de la presente divulgación, las condiciones adecuadas de hibridación con alta exigencia incluyen aquellas descritas anteriormente con la excepción que la temperatura de hibridación aumenta, por ejemplo, hasta 60-65°C o 65-70°C.
[0181] Los ejemplos de procedimientos para detectar ADN genómico, que codifica un polipéptido de fusión SLC34A2-ROSIutilizando el polinucleótido enunciado en (a) o (b) anteriormente distinto de la hibridación in situ mencionada, son mancha southern, macha northern y dot blot. En estos procedimientos, el gen de fusión mencionado anteriormente se detecta mediante hibridación en condiciones de moderada a alta exigencia el polinucleótido enunciado en (a) o (b) anteriormente con una membrana, donde un extracto de ácido nucleico obtenido a partir del espécimen biológico mencionado anteriormente se ha transcrito. Cuando se utiliza el polinucleótido (a), se puede determinar que el ADN genómico que codifica un polipéptido de fusión SLC34A2-ROS1 se ha detectado, cuando un polipéptido, que hibrida con un polinucleótido, que codifica la proteína ROS1, reconoce la misma banda desarrollada sobre la membrana.
[0182] El análisis de microarray genómico y el análisis de microarray de ADN son procedimientos adicionales para detectar el ADN genómico, que codifica un polipéptido de fusión SLC34A2-ROS1 utilizando el polinucleótido (b) mencionado anteriormente. En estos procedimientos, una matriz de polinucleótidos (b) se fija en un sustrato y el ADN genómico relevante se determina colocando el espécimen biológico en contacto con el polinucleótidos sobre la matriz. En PCR o secuenciación, el polinucleótido (c) enunciado a continuación se puede utilizar para amplificar de manera específica una parte o todo de un nucleótido de fusión SLC34A2-ROS1, utilizando ADN (ADN genómico, ADNc) o a Rn preparados a partir del espécimen biológico como una plantilla. (c) Un polinucleótido, que es un par de cebadores diseñados para atrapar un sitio de fusión de un polinucleótido, que codifica la proteína SLC34A2, y un polinucleótido, que codifica la proteína ROS1. El “polinucleótido que es un par de cebadores” es un primer conjunto, donde un cebador hibrida con un polinucleótido, que codifica la proteína SLC34A2, y el otro cebador hibrida con un polinucleótido, que codifica la proteína ROS1, en una secuencia de bases, tal como, el polinucleótido de fusión mencionado anteriormente que sirve como una diana. La longitud de estos polinucleótidos es normalmente de 15 a 100 bases y, preferiblemente, de 17 a 30 bases.
[0183] Desde los puntos de vista de precisión y sensibilidad de detección mediante PCR, el polinucleótido enunciado en (c) de la presente divulgación es preferiblemente una secuencia complementaria a la secuencia de bases del polinucleótido de fusión mencionado anteriormente a menos de 5000 bases desde el sitio de fusión del polinucleótido, que codifica la proteína SLC34A2, y el polinucleótido, que codifica la proteína ROS1.
[0184] Se puede diseñar de manera apropiada el "polinucleótido que es un par de cebadores" utilizando técnicas conocidas basadas en la secuencia de bases del polinucleótido de fusión SLC34A2-ROS1 que sirve como diana. Los ejemplos ventajosos del “polinucleótido que es un par de cebadores” son conjuntos de cebadores hechos de un cebador seleccionado del grupo, que consiste en SLC34A2-ROS1-F1, SLC34A2-int15-F1, SLC34A2-int15-F2, SLC34A2-ex16-F1, SLC34A2-ex23-F1, SLC34A2-ex24- F1, SLC34A2-F-orf2438 y SLC34A2-int15-F3.5, y un cebador seleccionado del grupo, que consiste en SLC34A2-ROS1- R1, ROS1-int11-R3, ROS1-int7-R1, ROS1-int11-R0.5, ROS1-int11-R1, ROS1-int7-R2 y ROS1-R-orf2364. Más preferiblemente, es SLC34A2-ROS1-F1 y SLC34A2-ROS1-R1, SLC34A2-int15-F1 y SLC34A2-ROS1-R1, SLC34A2-int15- F2 y ROS1-int11-R3, SLC34A2-ex16-F1 y SLC34A2-ROS1-R1, SLC34A2-ex23-F1 y SLC34A2-ROS1-R1, o cebador SLC34A2-ex24-F1 y cebador ROS1-int7-R1.
[0185] En algunas realizaciones, la presente divulgación da a conocer procedimientos para: identificar, evaluar o detectar una fusión SLC34A2-ROS1; procedimientos para identificar, valorar, evaluar y/o tratar un sujeto que tiene cáncer, por ejemplo, cáncer que tiene una fusión SLC34A2-ROS1, CD74-ROS1 o FIG-ROS1;moléculas de ácido nucleico de SLC34A2-ROS1, CD74-ROS1 o FIG-ROS1 aisladas, construcciones de ácido nucleico, células huésped que contienen las moléculas de ácido nucleico; polipéptidos SLC34A2-ROS1, CD74-ROS1, o FIG-ROS1 purificados y agentes de unión; reactivos de detección (por ejemplo, sondas, cebadores, anticuerpos, equipos capaces de, por ejemplo, detección específica de ácidos nucleicos o proteínas SLC34A2-ROS1, CD74-ROS1 o FIG-ROS1); ensayos de cribado para identificar moléculas que interactúan con, por ejemplo, inhiben, fusiones 5'SLC34A2-3'ROS1, 5'CD74-3'ROS1, o 5'FIG-3'ROS1, por ejemplo, nuevos inhibidores de quinasa; así como, ensayos y equipos para evaluar, identificar, valor y/o tratar un sujeto, que tiene cáncer, por ejemplo, un cáncer, que tiene uno o más de una fusión SLC34A2-ROS1, CD74-ROS1 o FIG-ROS1. Las composiciones y los procedimientos identificados en el presente documento se pueden utilizar, por ejemplo, para identificar nuevos inhibidores de SLC34A2- ROS1, CD74-ROS1, o FIG-ROS1; para evaluar, identificar o seleccionar sujeto, por ejemplo, un paciente, que tiene cáncer; y para tratar o prevenir un cáncer.
[0186] Las Moléculas de Ácido Nucleico SLC34A2-ROS1, CD74-ROS1 y FIG-ROS1. En un aspecto, la presente divulgación incluye una molécula de ácido nucleico (por ejemplo, una molécula de ácido nucleico aislada o purificada), que incluye un fragmento de un gen SLC34A2 o CD74 o FIG y un fragmento de un protooncogén ROS1. En una realización de la presente divulgación, la molécula de ácido nucleico incluye una fusión, por ejemplo, una fusión dentro de marco, o al menos un exón de SLC34A2, CD74 o FIG y un exón de ROS1 (por ejemplo, uno o más exones, que codifican un dominio tirosina quinasa ROS1 o un fragmento del mismo).
[0187] En una realización ilustrativa de la presente divulgación, la molécula de ácido nucleico 5'SLC34A2-3'ROS1 comprende una secuencia de SLC34A2 suficiente y ROS1 suficiente, de tal modo que la fusión 5'SLC34A2-3'ROS1 codificada tiene actividad quinasa constitutiva, por ejemplo, tiene actividad elevada, por ejemplo, actividad quinasa, en comparación con ROS1 de tipo salvaje, por ejemplo, en una célula de un cáncer al que se hace referencia en el presente documento. En una realización, la fusión 5'SLC34A2-3'ROS1 codificada comprende al menos 1, 2, 3, 4, 5, 6, 7, 9, 10 o 11 exones de SLC34A2 y al menos 1, 2, 3, 4, 5, 6, 7, 9,10, 11, 12 o 13 exones de ROS1. En una realización,
el polipéptido de fusión 5'SLC34A2-3'ROS1 codificada incluye un dominio quinasa helicoidal o un fragmento funcional del mismo y un dominio tirosina quinasa de ROS1 o un fragmento funcional del mismo.
[0188] En una realización ilustrativa, la molécula de ácido nucleico 5'CD74-3'ROS1 comprende una secuencia de CD74 suficiente y ROS1 suficiente, de tal modo que la fusión 5'CD74-3'ROS1 codificada tiene actividad quinasa constitutiva, por ejemplo, tiene actividad elevada, por ejemplo, actividad quinasa, en comparación con ROS1 de tipo salvaje, por ejemplo, en una célula de un cáncer al que se hace referencia en el presente documento. En una realización, la fusión 5'CD74-3'ROS1 codificada comprende al menos 1, 2, 3, 4, 5, 6 exones de CD74 y al menos 1,2, 3, 4, 5, 6, 7, 9,10, 11, 12 o 13 exones de ROS1. En una realización de la presente divulgación, el polipéptido de fusión 5'CD74-3'ROS1 codificada incluye un helicoidal; un dominio de proteína de membrana de ancla única del tipo II o un fragmento funcional del mismo y un dominio tirosina quinasa de ROS1 o un fragmento funcional del mismo.
[0189] En una realización ilustrativa de la presente divulgación, la molécula de ácido nucleico 5'FIG-3'ROS1 comprende una secuencia de FIG suficiente y ROS1 suficiente, de tal modo que la fusión 5'FIG-3'ROS1 codificada tiene actividad quinasa constitutiva, por ejemplo, tiene actividad elevada, por ejemplo, actividad quinasa, en comparación con ROS1 de tipo salvaje, por ejemplo, en una célula de un cáncer al que se hace referencia en el presente documento. En una realización de la presente divulgación, la fusión 5'FIG-3'ROS1 codificada comprende al menos 1, 1, 2, 3, 4, 5, 6, 7 o 8 exones de FIG y al menos 1, 1, 2, 3, 4, 5, 6, 7, 10, 11, 12 o 13 exones de ROS1. En una realización de la presente divulgación, el polipéptido de fusión 5'FIG-3'ROS1 codificada incluye un dominio de bobina enrollada o un fragmento funcional del mismo y un dominio tirosina quinasa de ROS1 o un fragmento funcional del mismo.
[0190] En una realización de la presente divulgación, la molécula de ácido nucleico de fusión SLC34A2-ROS1 incluyen una secuencia de nucleótidos, que tiene una fusión de marco de exón 2 y/o 4 de SLC34A2 con exón 32 y/o 34, y/o 35 de ROS1. En otra realización de la presente divulgación, la molécula de ácido nucleico incluye una secuencia de nucleótidos que incluye un punto de rotura. Por ejemplo, la fusión SLC34A2-ROS1 puede incluir una fusión de marco de al menos exón 4 de SLC34A2 o un fragmento del mismo (por ejemplo, exones 1-4 de SLC34A2 o un fragmento de los mismos) con al menos exón 32 y/o 34 de ROS1 o un fragmento del mismo (por ejemplo, exones 30-43 de ROS1 o un fragmento de los mismos). En algunas realizaciones de la presente divulgación, la fusión SLC34A2-ROS1 está en una configuración de 5'-SLC34A2 a 3'-ROS1. En una realización de la presente divulgación, la molécula de ácido nucleico incluye la secuencia de nucleótidos de exones 1-4 del gen SLC34A2 o un fragmento de la misma. En otra realización de la presente divulgación, la molécula de ácido nucleico incluye la secuencia de nucleótidos de exones 30-43, por ejemplo, 32 y/o 34, y/o 35 del gen ROS1 o un fragmento de la misma o una secuencia sustancialmente idéntica a la misma.
[0191] En una realización de la presente divulgación, la molécula de ácido nucleico de fusión CD74-ROS1 incluyen una secuencia de nucleótidos, que tiene una fusión de marco de exones 1-6 de CD74 con exón 32 y/o 34 de ROS1. En otra realización de la presente divulgación, la molécula de ácido nucleico incluye una secuencia de nucleótidos que incluye un punto de rotura. Por ejemplo, la fusión CD74-ROS1 puede incluir una fusión de marco de al menos exón 6 de CD74 o un fragmento del mismo (por ejemplo, exones 1-6 de CD74 o un fragmento de los mismos) con al menos exón 32 y/o 34 y/o 35 de ROS1 o un fragmento del mismo (por ejemplo, exones 30-43 de ROS1 o un fragmento de los mismos). En algunas realizaciones de la presente divulgación, la fusión CD74-ROS1 está en una configuración de 5'-CD74 a 3'-ROS1. En una realización de la presente divulgación, la molécula de ácido nucleico incluye la secuencia de nucleótidos de exones 1-6 del gen CD74 o un fragmento de la misma. En otra realización de la presente divulgación, la molécula de ácido nucleico incluye la secuencia de nucleótidos de exones 30-43, por ejemplo, 32 y/o 34, y/o 35 del gen ROS1 o un fragmento de la misma o una secuencia sustancialmente idéntica a la misma.
[0192] En una realización de la presente divulgación, la molécula de ácido nucleico de fusión FIG-ROS1 incluyen una secuencia de nucleótidos, que tiene una fusión de marco de exones 1-8, por ejemplo, 1-4 o 1-8 de FIG con exón 32 y/o 34, y/o 35 de ROS1. En otra realización de la presente divulgación, la molécula de ácido nucleico incluye una secuencia de nucleótidos que incluye un punto de rotura. Por ejemplo, la fusión FIG-ROS1 puede incluir una fusión de marco de al menos exón 4 y/o exón 8 de FIG o un fragmento del mismo (por ejemplo, exones 1-4 o 1-8 de FIG o un fragmento de los mismos) con al menos exón 32 y/o 34 y/o 35 de ROS1 o un fragmento del mismo (por ejemplo, exones 30-43 de ROS1 o un fragmento de los mismos). En algunas realizaciones de la presente divulgación, la fusión FIG-ROS1 está en una configuración de 5'-CD74 a 3'-ROS1. En una realización, la molécula de ácido nucleico incluye la secuencia de nucleótidos de exones 1-6 del gen CD74 o un fragmento de la misma. En otra realización de la presente divulgación, la molécula de ácido nucleico incluye la secuencia de nucleótidos de exones 30-43, por ejemplo, 32 y/o 34, y/o 35 del gen ROS1 o un fragmento de la misma o una secuencia sustancialmente idéntica a la misma.
[0193] En otras realizaciones de la presente divulgación, la molécula de ácido nucleico de fusión SLC34A2-ROS1 incluye una secuencia de nucleótidos, que codifica un polipéptido de fusión SLC34A2-ROS1, que incluye un fragmento de un gen SLC34A2 y un fragmento de un protooncogén ROS1. En una realización de la presente divulgación, la secuencia de nucleótidos, que codifica un polipéptido de fusión SLC34A2-ROS1, que incluye un dominio helicoidal o un fragmento funcional del mismo y un dominio tirosina quinasa de ROS1 o un fragmento funcional del mismo.
[0194] En otra realización de la presente divulgación, la molécula de ácido nucleico de fusión CD74-ROS1 incluye una
fusión CD74-ROS1, que incluye un cruce de fusión ente la transcripción ROS1 y la transcripción CD74. En otra realización de la presente divulgación, la molécula de ácido nucleico incluye una fusión, por ejemplo, una fusión de marco, de al menos exón 32 o 34, o 35 de ROS1 o un fragmento del mismo (por ejemplo, exones 30-43 de ROS1 o un fragmento de los mismos) y al menos exón 1 o un fragmento del mismo (por ejemplo, exones 1-6 de CD74 o un fragmento de los mismos). En algunas realizaciones de la presente divulgación, la fusión CD74-ROS1 está en una configuración de 5'-CD74 a 3'-ROS1. En una realización de la presente divulgación, la molécula de ácido nucleico incluye los nucleótidos, que corresponden a exones 30-43 de un gen ROS1, por ejemplo, exones 32, 34 o 35 o un fragmento de los mismos o una secuencia sustancialmente idéntica a los mismos.
[0195] En otra realización de la presente divulgación, la molécula de ácido nucleico de fusión FIG-ROS1 incluye una fusión FIG-ROS1, que incluye un cruce de fusión ente la transcripción ROS1 y la transcripción FIG. En otra realización de la presente divulgación, la molécula de ácido nucleico incluye una fusión, por ejemplo, una fusión de marco, de al menos exón 32 o 34, o 35 de ROS1 o un fragmento del mismo (por ejemplo, exones 30-43 de ROS1 o un fragmento de los mismos) y al menos exón 1 o 8 o un fragmento del mismo (por ejemplo, exones 1-8 de FIG o un fragmento de los mismos). En algunas realizaciones de la presente divulgación, la fusión FIG-ROS1 está en una configuración de 5'- FIG a 3'-ROS1. En una realización de la presente divulgación, la molécula de ácido nucleico incluye los nucleótidos, que corresponden a exones 30-43 de un gen ROS1, por ejemplo, exones 32, 34 o 35 o un fragmento de los mismos o una secuencia sustancialmente idéntica a los mismos.
[0196] En un aspecto relacionado, la presente divulgación incluye construcciones de ácido nucleico, que incluyen las moléculas de ácido nucleico de fusión SLC34A2-ROS1, CD74-ROS1 y FIG-ROS1 descritas en el presente documento. En algunas realizaciones de la presente divulgación, las moléculas de ácido nucleico se unen de manera operativa con una secuencia reguladora nativa o heteróloga. Se incluyen también vectores y células huésped, que incluyen las moléculas de ácido nucleico SLC34A2-ROS1 descritas en el presente documento, por ejemplo, vectores y células huésped adecuados para producir las moléculas de ácido nucleico y polipéptidos descritos en el presente documento.
[0197] En otro aspecto, la presente divulgación incluye moléculas de ácido nucleico, que reduce o inhibe la expresión de una molécula de ácido nucleico, que codifica una fusión SLC34A2-ROS1 descrita en el presente documento. Los ejemplos de tales moléculas de ácido nucleico incluyen, por ejemplo, moléculas antisentido, ribozimas, ARNi, moléculas con triple hélice, que hibridan con un ácido nucleico, que codifica SLC34A2-ROS1, CD74-ROS1 y FIG-ROS1 o una región reguladora de transcripción de SLC34A2-ROS1, CD74-ROS1 y FIG-ROS1 y bloquea o reduce expresión de ARNm de SLC34A2-ROS1, CD74-ROS1 y FIG-ROS1.
[0198] La presente divulgación incluye también una molécula de ácido nucleico, por ejemplo, fragmento de ácido nucleico, apropiado como sonda, cebador, cebo o miembro de biblioteca, que incluye, flanquea, hibrida con, que es útil para identificar o se basa de otro modo en las fusiones ROS 1 descritas en el presente documento. En algunas realizaciones de la presente divulgación, la sonda, el cebador o el cebo es un oligonucleótido, que permite capturar, detectar o aislar una molécula de ácido nucleico de fusión ROS1 descrita en el presente documento. El oligonucleótido puede comprender una secuencia de nucleótidos sustancialmente complementaria con un fragmento de las moléculas de ácido nucleico de fusión ROS1 descritas en el presente documento. La identidad de secuencia entre el fragmento de ácido nucleico, por ejemplo, el oligonucleótido, y la secuencia diana de fusión ROS1 no tiene que ser exacta, siempre y cuando las secuencias son suficientemente complementarias para permitir capturar, detectar o aislar la secuencia diana. En una realización, el fragmento de ácido nucleico es una sonda o un cebador, que incluye un oligonucleótido entre aproximadamente 5 y 25, por ejemplo, entre 10 y 20, 10 y 15 nucleótidos de longitud. En otras realizaciones, el fragmento de ácido nucleico es un cebo, que incluye un oligonucleótido entre aproximadamente 100 hasta 300 nucleótidos, 130 hasta 230 nucleótidos, 150 hasta 200 nucleótidos, 200 hasta 350, 350 hasta 950, 300 hasta 600, 500-1000, 750-2000 nucleótidos de longitud y no incluye necesariamente los ácidos nucleicos de fusión ROS1.
[0199] En una realización de la presente divulgación, el fragmento de ácido nucleico se puede utilizar para identificar o capturar, por ejemplo, mediante hibridación, una fusión SLC34A2-ROS1 o una fusión CD74-ROS1, o una fusión FIG-ROS1. En un ejemplo ilustrativo, el fragmento de ácido nucleico puede ser una sonda, un cebador para su uso en identificación o captura, por ejemplo, mediante hibridación condiciones de moderada o alta exigencia, de una fusión SLC34A2-ROS1 o una fusión CD74-ROS1, o una fusión FIG-ROS1 descritas en el presente documento. En una realización de la presente invención, se puede utilizar el fragmento de ácido nucleico para identificar o capturar un punto de rotura SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS 1. En una realización de ejemplo de la presente divulgación, el fragmento de ácido nucleico hibrida con una secuencia de nucleótidos dentro de una reorganización cromosómica, que crea una fusión de marco de exón 4 o 13 de SLC34A2 con exón 32, 34 o 35 de ROS1 (por ejemplo, una secuencia dentro de una translocación en cromosoma 6).
[0200] Se pueden utilizar las sondas o los cebadores descritos en el presente documento, por ejemplo, para la detección FISH o la amplificación PCR, o para la confirmación RT-PCT de reorganizaciones ROS1 y la identificación de socios de unión. En una realización de ejemplo de la presente divulgación, donde la detección se basa en amplificación PCR de los SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1, se puede realizar los cruces de fusión utilizando un cebador o un par de cebadores, por ejemplo, para amplificar una secuencia, que flanquea las cruces de fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1 descritas en el presente documento, por ejemplo, las mutaciones o el cruce de una reorganización cromosómica descrita en el presente documento. En una realización de la presente
divulgación, un par de cebadores de oligonucleótidos aislados pueden amplificar una región, que contiene o es adyacente a una posición en la fusión SLC34A2-ROS1. Por ejemplo, se pueden asignar los cebadores avanzados para hibridar con una secuencia de nucleótidos dentro de una secuencia de SLC34A2, CD74, o FIG genómico o de ARNm. En diferentes realizaciones de la presente divulgación, los cebadores para ROS1, SLC34A2 y FIG (diferentes especies incluyendo los humanos) están disponibles comercialmente en Qiagen (Números Cat. QF0018545, q F00449078 y QF00278992, Qiagen, Gaithersburg, MD, EEUU). En otras realizaciones de la presente divulgación, la confirmación de la presencia de polinucleótidos de fusión ROS1 se puede llevar a cabo en muestras tumorales de paciente utilizando un panel de cebadores de PCR, que incluyen cebadores avanzados de exón 4 de SLC34A2 y exón 6 de CD74 cada uno emparejado con un cebador reverso de exón 32 y exón 34 de ROS1. Se puede extraer el ARN de la muestra tumoral de tejido FFPE utilizando el procedimiento de Agencourt Formapure (Agencourt Biosciences, Beverly, MA, EEUU) y de transcripción reversa utilizando el kit de síntesis de ADNc Superscript III disponible comercialmente en Invitrogen, Carlsbad, CA, EEUU. Se puede utilizar los siguientes cebadores para determinar puntos de rotura de SLC34A2-ROS1 o CD74- ROS1: exón 4 avanzado de SLC34A2, TCGGATTTCTCTACTTTTTCGTG (SEQ ID NO:3); exón 6 avanzado de CD74, CTCCTGTTTGAAATGAGCAGG (SEQ ID NO:4); exón 32 reverso de ROS1, GGAATGCCTGGTTTATTTGG (SEQ ID NO:5); y exón 34 reverso de ROS1, TGAAACTTGTTTCTGGTATCCAA (SEQ ID NO:6). Se pueden llevar a cabo las amplificaciones de PCR en cualquier máquina cicladora térmica de PCR, por ejemplo, un Gradiente deEppendorf Mastercycler (Eppendorf, Hamburg, Alemania), utilizando polimerasa con Etiqueta de Platino (Invitrogen) en condiciones estándares con 40 ng de ADNc. ADNc se secuenció utilizando el kit BigDye 3.0 (Life Technologies, Carlsbad, CA).
[0201] El fragmento de ácido nucleico se puede etiquetar de manera detectable con, por ejemplo, un radiomarcado, una etiqueta fluorescente, una etiqueta bioluminiscente, una etiqueta quimioluminiscente, una etiqueta de enzima, una etiqueta de pareja de unión o se puede incluir una etiqueta de afinidad; una etiqueta o identificador (por ejemplo, un adaptador, un código de barras u otro identificador de secuencia).
[0202] en otra realización de ejemplo de la presente divulgación, se da a conocer un procedimiento para determinar la presencia de una fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1, que comprende: adquirir conocimiento directamente de que una molécula o un polipéptido de SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1 está presente en una muestra a partir de un sujeto. La muestra puede ser una muestra compuesta por fluido, células, tejido, por ejemplo, un tejido tumoral, una muestra de proteína, una biopsia tumoral o una célula de tumor circulante o ácido nucleico, puede incluir una muestra de ácido nucleico, se puede escoger de un cáncer de pulmón, que incluye un NSCLC, un SCLC, un SCC o una combinación de los mismos y un adenocarcinoma o un melanoma.
[0203] Se puede detectar la fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1 en una molécula de ácido nucleico utilizando cualquiera de los siguientes procedimientos: ensayo de hibridación de ácido nucleico, ensayos basados en amplificación, ensayo PCR-RFLP, PCR en tiempo real, secuenciación, análisis de cribado, FISH, cariotipado espectral o MFISH, hibridación genómica comparativa), hibridación in situ, SSP, HPLC o genotipado de espectrometría de masas.
[0204] Se describe un procedimiento para detectar un polipéptido o una proteína de fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1 incluyendo el procedimiento de contacto de una muestra de proteína con un reactivo, que se une de manera específica con un polipéptido de fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS 1; y detectando la formación de un complejo del polipéptido de fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1 y el reactivo. Los procedimientos para detectar polipéptido incluyen un reactivo etiquetado con un grupo detectable para facilitar la detección del enlace y el reactivo suelto, donde el reactivo es una molécula de anticuerpo, donde se evalúa el nivel o la actividad de fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1, donde se detecta la fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1 antes de iniciar, durante o después de un tratamiento en un sujeto, donde se detecta la fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1 en el momento de diagnóstico con un cáncer, donde se detecta la fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1 en un intervalo predeterminado, por ejemplo, un primer momento y al menos en un momento posterior.
[0205] También se incluyen en la presente divulgación las respuestas a tratamiento, especialmente, cuando, sensible a una determinación de la presencia de la fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1, se utiliza uno o más de los siguientes: (1) estratificar una población de pacientes; (2) identificar o seleccionar el sujeto como probable o improbable a responder a un tratamiento; (3) seleccionar una opción de tratamiento; y/o (4) pronosticar el curso temporal de la enfermedad en el sujeto.
[0206] Se puede utilizar una molécula de ácido nucleico asilada o purificada, que codifica una fusión o un punto de rotura de SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1, que comprende un fragmento del mismo, con fines confirmatorios y para tratamiento específico directo de NSCLC y cáncer positivo en la fusión de ROS1 de adenocarcinoma de pulmón utilizando los compuestos de la presente invención. Se puede utilizar otras construcciones para el tratamiento específico directo de cánceres positivo en la fusión de ROS1, por ejemplo, NSCLC con ROS1 reorganizado, incluyendo: una molécula de ácido nucleico de fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1 nucleica aislada o purificada unida de manera operativa con una secuencia reguladora nativa o heteróloga. Un vector aislado o purificado, que comprende una molécula de ácido nucleico, que codifica una fusión o un punto de rotura de SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1, que comprende un fragmento de la misma. Una célula huésped que
comprende un vector. Una molécula de ácido nucleico, que reduce o inhibe de manera específica la expresión de una molécula de ácido nucleico, que codifica una fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1, que se puede seleccionar de una molécula antisentido, ribozima, siARN o molécula de triple hélice. Un polipéptido de fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1 aislado o purificado o punto de rotura, que contiene un fragmento del mismo. El polipéptido de fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1 aislado o purificado, que tiene actividad quinasa ROS1 y/o una actividad de dimerización o multidimerización. Una molécula de anticuerpo aislada o purificada, que une de manera específica un polipéptido de fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1. Una molécula de anticuerpo, donde dicha molécula de anticuerpo es una molécula de anticuerpo específico con el polipéptido de fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1
[0207] Los ejemplos del procedimiento para detectar el producto de traducción del polinucleótido de fusión SLC34A2-ROS1, o CD74-ROS1, o FIG- ROS1 en la presente divulgación son inmunotinción, transferencia western, ELISA, citometría de flujo, inmunoprecipitación y análisis de matriz de anticuerpos. En estos procedimientos, se utiliza un anticuerpo que se une con un polipéptido de fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1. Los ejemplos de tal anticuerpo son un anticuerpo específico con un polipéptido, que contiene un sitio de fusión de la proteína SLC34A2-ROS1, o CD74-ROS1, o FIG y la proteína ROS1 (al que también se refiere como “anticuerpo con sitio de fusión específico” de aquí en adelante), un anticuerpo, que se une con un polipéptido hecho de una región en el lado C terminal a partir del sitio de fusión mencionado anteriormente de la proteína (al que se refiere como “anticuerpo con terminal ROS1-C” de aquí en adelante) y un anticuerpo, que se une con un polipéptido hecho de una región en el lado N terminal a partir del sitio de fusión mencionado anteriormente de la proteína SLC34A2, o CD74, o FIG (a la que se refiere como “anticuerpo N-terminal de SLC34A2, o CD74, o FIG” de aquí en adelante). Aquí, “anticuerpo de sitio de fusión específico” significa un anticuerpo que se une de manera específica con un polipéptido, que contiene el sitio de fusión mencionado anteriormente, pero no se une ni con proteína SLC34A2, o CD74, o FIG de tipo salvaje (tipo normal) o proteína ROS1 de tipo salvaje (tipo normal).
[0208] Se puede detectar un polipéptido de fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1 mediante el anticuerpo con sitio de fusión específico mencionado anteriormente o una combinación del anticuerpo ROS1-C terminal mencionado anteriormente y anticuerpo SLC34A2, o CD74, o FIG -N terminal. Sin embargo, como no se detecta casi ninguna expresión de la proteína ROS1, por ejemplo, en células de pulmón normales, se puede detectar la presencia de un polipéptido de fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1 en tejido de adenocarcinoma de pulmón, incluso si el anticuerpo ROS1-C terminal en sí se utiliza en inmunotinción.
[0209] En diferentes realizaciones, se pueden llevar a cabo las aplicaciones de tinción inmunohistoquímica (IHC) y transferencia western para la identificación de polipéptidos de fusión ROS1 de la presente divulgación utilizando anticuerpos fosfo-ROS1, ROS1, que están disponibles comercialmente en Cell Signaling Technology (Danvers, MA, EEUU). En una realización, se puede llevar a cabo la identificación de células cancerígenas positivo en la fusión de ROS1, por ejemplo, NSCLC con células de cáncer de pulmón reorganizadas en ROS1 con anticuerpo anti-ROS1 monoclonal D4D6 de conejo, Número Cat. 3287 (Cell Signaling Technology, Danvers, MA, EEUU).
[0210] En algunas realizaciones, se puede preparar el "anticuerpo que se une con un polipéptido de fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1" por un experto en la técnica seleccionando un procedimiento apropiado conocido. Un ejemplo de tales procedimientos conocidos es un procedimiento, donde un animal inmune es inoculado con el polipéptido mencionado anteriormente hecho de una porción C terminal de la proteína ROS1, un polipéptido de fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1, el polipéptido mencionado anteriormente hecho de una porción N terminal de la proteína SLC34A2 o CD74, o FIG, etc., activando de este modo el sistema inmune del animal, y, a continuación, se recupera el suero de sangre del animal (anticuerpo policlonal), y los procedimientos para producir anticuerpos monoclonales, tales como, el procedimiento de hibridoma, el procedimiento de ADN recombinante y la técnica de phage display. Si se utiliza un anticuerpo al que se une una sustancia etiquetada, se puede detectar directamente la proteína diana detectando la etiqueta. La sustancia etiquetada no está limitada particularmente, siempre y cuando puede unirse con el anticuerpo y se puede detectar. Los ejemplos incluyen peroxidasa, p-D-galactosidasa, microperoxidasa, peroxidasa de rábano (PHR), isotiocianato de fluorescencia (FITC), isotiocianato de rodamina (RITC), fosfatasa alcalina, biotina y sustancias radioactivas. Además, adicionalmente a los procedimientos que detectan directamente una proteína diana utilizando un anticuerpo con el que se une una sustancia etiquetada, se pueden utilizar los procedimientos que detectan de manera indirecta la proteína diana utilizando proteína G, proteína A o un anticuerpo secundario con el que se une una sustancia etiquetada.
[0211] Si se detecta la presencia de un polipéptido o polinucleótido de fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1 en un espécimen aislado de un sujeto utilizando los procedimientos mencionados anteriormente, cabe pensar que la eficacia de tratamiento de cáncer utilizando un inhibidor de tirosina quinasa ROS1, tal como, un compuesto de la Fórmula I, Ia o el compuesto 1 o una sal farmacéuticamente aceptable del mismo, será alta en aquel paciente, mientras que si no se detecta SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1, cabe pensar que la eficacia de tratamiento de cáncer utilizando un inhibidor de tirosina quinasa ROS1 será baja en aquel paciente.
[0212] Como se describe anteriormente, cualquiera de los polinucleótidos enunciados en (a) hasta (c) a continuación, que tiene una longitud de cadena de al menos 15 bases, se puede utilizar en la detección de la presencia o la ausencia de un polipéptido de fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1;
(a) un polinucleótido, que es al menos una sonda seleccionada del grupo, que consiste en sondas, que hibridan con un polinucleótido, que codifica la proteína SLC34A2, o CD74, o FIG y las sondas, que hibridan con un polinucleótido, que codifica la proteína ROS1;
(b) un polinucleótido, que es una sonda, que hibrida con un sitio de fusión de un polinucleótido, que codifica la proteína SLC34A2, o CD74, o FiG, y un polinucleótido, que codifica la proteína ROS1;
(c) un polinucleótido, que es un par de cebadores diseñados para atrapar un sitio de fusión de un polinucleótido, que codifica la proteína SLC34A2, o CD74, o FIG, y un polinucleótido, que codifica la proteína ROS1.
[0213] Estos polinucleótidos tienen una secuencia de bases complementaria con la secuencia de bases específica del gen diana. En el presente documento, “complementario” puede significar no completamente complementario, debido a que hibrida, por ejemplo, bajo condiciones de exigencia moderada hasta alta. Por ejemplo, estos polipéptidos tienen 80% o más, preferiblemente 90% o más, más preferiblemente 95% o más y más preferiblemente 100% de homología con la secuencia de bases específica.
[0214] En los polinucleótidos de (a) hasta (c), en parte o todos ADN o ARN, nucleótidos se pueden sustituir con ácidos nucleicos artificiales, tales como, PNA (ácido nucleico de poliamida, ácido nucleico de péptido), ANB (marca registrada, ácido nucleico bloqueado, ácido nucleico cerrado), ENA (marca registrada, ácidos nucleicos de 2'-O, 4'-C-etilenocerrados), GNA (ácido nucleico de glicerol) y ATN (ácido nucleico treósico).
[0215] Además, como se describe en el presente documento, un anticuerpo, que se une a un polipéptido de fusión SLC34A2-ROS1, o CD74-ROS1, o FIG-ROS1 se usa de manera ventajosa para detectar el producto de traducción de un polinucleótido de fusión SLC34A2-ROS1, o CD74-ROS1, o FIG- r Os 1. Por consiguiente, la presente divulgación proporciona un fármaco para determinar la efectividad de tratamiento de cáncer utilizando un inhibidor de tirosina quinasa ROS1, que comprende este anticuerpo.
[0216] El procedimiento de detección de un gen de fusión de la presente divulgación incluye una etapa para detectar la presencia de polinucleótidos de la presente especificación en una muestra obtenida de un sujeto de prueba. Igual que la muestra obtenida de un sujeto de prueba, se utilizan las sustancias recogidas de un sujeto de prueba (muestras aisladas de un cuerpo vivo), específicamente, cualquier tipo de fluido corporal (preferiblemente sangre) recogido, lavados alveolares y bronquiales, muestras, que se han sometido a biopsia y muestras de flema. Preferiblemente, se utiliza una muestra de biopsia o una muestra de flema de un área afectado en el pulmón de un sujeto de prueba. A partir de la muestra, se puede extraer y usar ADN genómico. Adicionalmente, se pueden utilizar las transcripciones del mismo (productos producidos como un resultado de transcripción o traducción de un genoma; por ejemplo, ARNm, ADNc y proteínas). Particularmente, es preferible preparar y utilizar ARNm o ADNc.
[0217] El ADN genómico se puede extraer utilizando los procedimientos conocidos y la extracción se puede llevar a cabo fácilmente utilizando un equipo de extracción de ADN disponible comercialmente.
[0218] La etapa de detección se puede llevar a cabo según los procedimientos de análisis génico conocidos (por ejemplo, los procedimientos conocidos, que se usan de manera común como procedimientos de detección de genes, tales como PCR, LCR (Reacción en cadena de ligasa), SDA (Amplificación de desplazamiento en cadena), NASBA (Amplificación basada en secuencia de ácidos nucleicos), ICAN (Amplificación iniciada por cebador isotérmico y quimérico de ácidos nucleicos), procedimiento LAMP (Amplificación isotérmica mediada en lazo), procedimiento TMA (sistema TMA de sonda de genes), procedimiento de hibridación in situ y microarrays). Por ejemplo, una tecnología de hibridación, donde un ácido nucleico hibridado con un polinucleótido a detectar se utiliza como una sonda, una tecnología de amplificación de genes, donde el ADN hibridado con un polinucleótido a detectar se utiliza como un cebador o se utilizan los similares.
[0219] Especialmente, la detección se lleva a cabo utilizando los ácidos nucleicos derivados de una muestra obtenida de un sujeto de prueba, por ejemplo, ARNm y los similares. La cantidad de ARNm se mide utilizando un procedimiento de reacción de amplificación génica mediante cebadores que se designan de tal modo que son capaces de amplificar especialmente la secuencia de un polinucleótido detectado. Los cebadores utilizados en el procedimiento de detección de la presente divulgación o los cebadores incluidos en el equipo de detección no se limitan de manera particular, siempre y cuando los cebadores pueden amplificar de manera específica la secuencia de un polinucleótido a detectar y se diseñan basándose en la secuencia de bases de un polinucleótido a detectar. Los cebadores utilizados en el procedimiento de observación de amplificación por PCR utilizando software de diseño de cebadores (por ejemplo, Primer Express fabricado por PE Biosystems) y los similares. Adicionalmente, dado que mayor es el tamaño de un producto de PCR, más se empeora la eficacia de amplificación, es apropiado para un primer cebador y un cebador antisentido diseñarlos de tal modo que el tamaño de los productos de amplificación obtenidos durante la amplificación de ARNm o ADNc sea 1kb o menos.
[0220] Más específicamente, un cebador de sentido (5'-cebador) se diseña a partir de una porción, que codifica SLC34A2, y un cebador antisentido (3'-cebador) se diseña a partir de una porción, que codifica ROS1. Es preferible utilizar el cebador incluido en el equipo de detección de la presente divulgación, y es más preferible utilizar el cebador, que está incluido de manera más adecuado en el equipo de detección. En el procedimiento de observación de amplificación por PCR, también, es posible diseñar PCR múltiplex para detectar todos los polinucleótidos en un líquido
de reacción único, mezclando los cebadores sentido mencionados anteriormente, que corresponden a genes respectivos. Utilizando el procedimiento adecuado para cada tecnología de amplificación, es posible confirmar si un gen diana (el gen entero o una porción específica del mismo) se ha ampliado o no. Por ejemplo, en el procedimiento de PCR, los productos de PCR se analizan utilizando electroforesis en gel de agarosa y se someten a tinción con bromuro de etidio y los similares, por medio del cual es posible confirmar si los fragmentos amplificados, que tienen un tamaño deseado, se han obtenido o no. Cuando se han obtenidos los fragmentos amplificados, que tienen un tamaño deseado, esto indica que un polinucleótido a detectar está presente en la muestra obtenida de un sujeto de prueba. La presencia de un polinucleótido a detectar se puede detectar de esta manera.
[0221] El procedimiento de detección de un gen de fusión de la presente divulgación incluyen preferiblemente una etapa de detección de la presencia de un polinucleótido específico en una muestra obtenida de un sujeto de prueba utilizando una reacción de amplificación y una etapa de detección si los fragmentos amplificados, que tienen un tamaño deseado, se han obtenido o no.
[0222] La detección, que utiliza una tecnología de hibridación, se lleva a cabo utilizando, por ejemplo, hibridación northern, procedimiento de dot blot, procedimiento de microarray de ADN y procedimiento de protección de ARN. Cuando se utilizan sondas para la hibridación, es posible utilizar una sonda, que comprende secuencias, que consisten en 16 bases respectivamente más arriba o más abajo del punto de fusión como un centro de una molécula de ácido nucleico, que consiste en al menos 32 bases consecutivas, que hibrida con un polinucleótido a detectar o con una cadena complementaria del mismo en unas condiciones de exigencia moderada a alta (preferiblemente bajo una condición de exigencia alta) o comprende cadenas complementarias del mismo.
[0223] También, es posible utilizar una tecnología de amplificación génica, tal como, RT-PCR. En el procedimiento de RT-PCR, el procedimiento de observación (en tiempo real de PCR) de amplificación por PCR se lleva a cabo durante el proceso de amplificación génica, por medio del cual se puede analizar de manera más cuantitativa la presencia de un polinucleótido a detectar. Se pueden utilizar los procedimientos de observación de amplificación por PCR. PCR en tiempo real es un procedimiento conocido y se puede llevar a cabo fácilmente utilizando instrumentos y equipos disponibles comercialmente para este procedimiento.
[0224] El procedimiento de detección de una proteína de fusión en algunas realizaciones de la presente invención incluye una etapa de detección de la presencia de un polipéptido específico en una muestra obtenida de un sujeto de prueba, que es, un polipéptido específico codificado por un polinucleótido a detectar (de aquí en adelante, nombrado un polipéptido a detectar). Tal etapa de detección se puede llevar a cabo utilizando el procedimiento de inmunoensayo o el procedimiento de ensayo de actividad de enzimas, que se lleva a cabo mediante preparación de un líquido solubilizado derivado de una muestra obtenida de un sujeto de prueba (por ejemplo, un tejido o células cancerígenos obtenidos de un sujeto de prueba) y combinación de un polipéptido a detectar en el líquido con un anticuerpo anti-SLC34A2, o anti-CD74, o anti-FIG o un anticuerpo anti-ROS1. Preferiblemente, es posible utilizar técnicas que usan un anticuerpo monoclonal o un anticuerpo policlonal específico con un péptido a detectar, tal como, procedimiento de inmunoensayo de enzimas, procedimiento ELISA tipo sándwich de doble anticuerpo, procedimiento de inmunoensayo fluorescente, procedimiento de radioinmunoensayo y procedimiento de transferencia western.
[0225] Cuando el polinucleótido a detectar o el polipéptido a detectar en el procedimiento de detección de la presente divulgación es detectado a partir de una muestra obtenida de un sujeto de prueba, el sujeto de prueba es un sujeto (paciente), quien tiene cáncer con el polinucleótido positivo y se le proporcionará el tratamiento utilizando inhibidores de ROS1.
[0226] El equipo de detección de la presente divulgación comprende al menos cebadores de sentido y antisentido, que se designan de tal modo que son capaces amplificar de manera específica un polinucleótido a detectar en el procedimiento de detección de la presente divulgación El conjunto de cebadores sentido y antisentido es un conjunto de polinucleótidos, que funcionan como cebadores para amplificar un polinucleótido a detectar.
[0227] En una realización, el primer conjunto de la presente divulgación para la detección de un gen de fusión comprende (1) un conjunto de cebadores, que comprende un cebador sentido diseñado a partir de una porción, que codifica SLC34A2, o CD74, o FIG, y un cebador antisentido diseñado a partir de una porción, que codifica ROS1, y es para detectar un gen de fusión de gen SLC34A2, o CD74, o FIG y ROS1, donde el cebador antisentido consiste en una molécula de ácido nucleico (preferiblemente una molécula de ácido nucleico, que consiste en al menos 16 bases), que hibrida con un “polinucleótido a detectar” en condiciones de moderada a alta exigencia (preferiblemente en condiciones de alta exigencia), que hibrida con una cadena complementaria del “polinucleótido a detectar” en una condición rigurosa (preferiblemente en condiciones de alta exigencia).
[0228] Los siguientes conjuntos de cebadores (2) y (3) se incluyen en el primer conjunto como variantes más específicas del primer conjunto (1).
[0229] (2) Un conjunto de cebadores de un cebador sentido, que consiste en un oligonucleótido, que consiste en al menos cualquiera de 16 bases consecutivas.
[0230] (3) Un conjunto de cebadores de un cebador sentido, que consiste en un oligonucleótido, que consiste en al menos cualquiera de 16 bases consecutivas.
[0231] En estos conjuntos de cebadores (1) a (3), un intervalo entre las posiciones, donde el cebador sentido y el cebador antisentido se seleccionan, es preferiblemente 1kb o menos, o el tamaño de un producto de amplificación por el cebador sentido y el cebador antisentido es preferiblemente 1 kb o menos.
[0232] Adicionalmente, el cebador tiene una longitud de cadena, que consiste en 15 hasta 40 bases en general, preferiblemente, que consiste en 16 a24 bases, más preferiblemente, que consiste en 18 hasta 24 bases, y particularmente preferiblemente, que consiste en 20 hasta 24 bases.
[0233] El conjunto de cebadores se puede utilizar para amplificar y detectar un polinucleótido a detectar. Además, aunque no está limitado de manera particular, los cebadores respectivos incluidos en el conjunto de cebadores de la presente divulgación se pueden preparar utilizando, por ejemplo, síntesis química.
[0234] Cebadores ROS1, SLC34A2, o CD74 de ejemplo operables para detectar polinucleótidos de fusión ROS1 se dan a conocer en la Tabla 2.
Tabla 2
El conjunto de cebadores de ejemplo para identificar diferentes proteínas de fusión de ROS1 según la presente divulgación.
Nombre de cebador Secuencia de cebador (5'-3')
ROS1 InvPCR F1 GTCTGGCATAGAAGATTAAAG (SEQ ID NO: 7)
ROS1 InvPCR F2 AAACGAAGACAAAGAGTTGG (SEQ ID NO: 8)
ROS1 InvPCR R1 GTCAGTGGGATTGTAACAAC (SEQ ID NO: 9)
ROS1 Break Rev CAGCTCAGCCAACTCTTT (SEQ ID NO: 11)
ROS1 E43R1 TCTATTTCCCAAACAACGCTATTAATCAGACCC (SEQ ID NO: 12) ROS1 E34R TGAAACTTGTTTCTGGTATCCAA (SEQ ID NO: 13)
CD74 E5F CCTGAGACACCTTAAGAACACCA (SEQ ID NO: 14) SLC34A2 E4F TCGGATTTCTCTACTTTTTCGTG (SEQ ID NO: 15)
[0235] Un procedimiento de cribado de fármaco anticáncer incluye: contacto de un compuesto de muestra con una célula, que expresa la proteína de fusión; y medición del nivel de expresión de proteína de fusión en la célula, donde el nivel de expresión de proteína de fusión en la célula tratada con el mismo compuesto se disminuye en comparación con aquel de antes del tratamiento con el compuesto de muestra o aquel en una célula no tratada, el compuesto de muestra se determina como un compuesto candidato para el fármaco anticáncer.
[0236] El procedimiento de cribado de fármaco anticáncer puede incluir, además, la etapa de medición del nivel de expresión de proteína de fusión en la célula antes del tratamiento del mismo compuesto. En este caso el compuesto de muestra se puede determinar como un compuesto candidato para el fármaco anticáncer, donde el nivel de expresión de proteína de fusión después de tratamiento del compuesto de muestra se disminuye en comparación con aquel antes del tratamiento con el compuesto de muestra en la misma célula. Alternativamente, el procedimiento de cribado de un fármaco anticáncer puede incluir proporcionar células, que expresan la proteína de fusión, y contacto de un compuesto de muestra con una parte de las células proporcionadas. En este caso el compuesto de muestra se puede determinar como un compuesto candidato para el fármaco anticáncer, cuando el nivel de expresión de proteína de fusión en la célula contactada con el mismo compuesto se disminuye en comparación con aquel en las células, que no entran en contacto con el compuesto de muestra.
[0237] La célula utilizada en el procedimiento de cribado puede ser una célula derivada de una célula cancerígena, donde el gen de fusión o la proteína de fusión se expresa y/o se activa, un extracto de la célula o un cultivo de la célula. La célula cancerígena puede ser una célula cancerígena sólida, particularmente un cáncer de pulmón, por ejemplo, un cáncer de pulmón de células no pequeñas como un adenocarcinoma de pulmón, como se describe anteriormente.
[0238] Otra realización más de la presente divulgación proporciona un procedimiento de cribado de fármaco anticáncer contra cáncer de pulmón: tratando una célula, que expresa la proteína de fusión con un compuesto de muestra; midiendo el nivel de expresión de proteína de fusión en la célula, donde el nivel de expresión de proteína de fusión en la célula tratada con el compuesto de muestra disminuye en comparación con aquel antes del tratamiento con el compuesto de muestra o aquel en una célula no tratada, el compuesto de muestra se determina como un compuesto candidato para el fármaco anticáncer contra cáncer de pulmón.
[0239] Las proteínas de fusión ROS1 (es decir, SLC34A2-ROS1, CD74-ROS1 y FIG-ROS1) se pueden usar como un marcador para diagnosticar un cáncer de pulmón o para tratar o prevenir o tratar un cáncer de pulmón. El tratamiento
o la prevención de cáncer de pulmón comprende la etapa de administración de una cantidad terapéuticamente eficaz de al menos un inhibidor de proteína de fusión, al menos un inhibidor del gen de fusión, que codifica la proteína de fusión, al menos un inhibidor de gen, que codifica ROS1 o una combinación de los mismos a un paciente que lo necesita. El inhibidor puede ser, tal como, Fórmula I, Ia o compuesto 1 o una sal farmacéuticamente aceptable del mismo.
[0240] Otra realización de la presente divulgación proporciona un procedimiento de prevención y/o tratamiento de un cáncer, que comprende administra una cantidad terapéuticamente eficaz de al menos un inhibidor de la proteína de fusión, al menos un inhibidor del gen de fusión, que codifica la proteína de fusión, al menos un inhibidor de un gen, que codifica ROS1, o una combinación de los mismos a un paciente que lo necesita, cualquiera de los cuales puede ser los compuestos de la Fórmula 1 y los otros compuestos específicos descritos en el presente documento. Además, el procedimiento puede comprender la etapa de identificación del paciente, quien necesita la prevención y/o el tratamiento de un cáncer antes de la etapa de administración de tal tratamiento. Otra realización de la presente divulgación proporciona una composición para prevenir y/o tratar un cáncer, que comprende administrar al menos un inhibidor de la proteína de fusión, que incluye solamente la Fórmula 1 o con al menos un inhibidor del gen de fusión, que codifica la proteína de fusión, al menos un inhibidor de un gen, que codifica ROS1, o una combinación de los mismos. Otra realización de la presente divulgación proporciona un uso de un inhibidor de la proteína de fusión, un inhibidor del gen de fusión, que codifica la proteína de fusión, un inhibidor de un gen, que codifica ROS1 o una combinación de los mismos para prevenir y/o tratar un cáncer. El inhibidor puede ser, tal como, Fórmula I, Ia o compuesto 1 o una sal farmacéuticamente aceptable del mismo.
[0241] En la presente divulgación, el cáncer puede ser un cáncer de pulmón, en particular, un cáncer de pulmón de células pequeñas (SCLC) o un cáncer de pulmón de células no pequeñas (NSCLC), tal como un adenocarcinoma de pulmón, un carcinoma pulmonar de células escamosas o un carcinoma pulmonar de células grandes.
[0242] La composición, donde el inhibidor de la proteína de fusión ROS1 es al menos uno seleccionado del grupo, que consiste en un aptámero que se une de manera específica con la proteína de fusión, un anticuerpo, que se une de manera específica con la proteína de fusión y el inhibidor del gen de fusión o el gen, que codifica ROS1, es al menos uno seleccionado del grupo, que consiste en siARN, shARN, miARN y un aptámero, que es capaz de unirse de manera específica con el gen de fusión o el gen, que codifica ROS1.
[0243] Cualquier inhibidor de quinasa ROS1, que inhibe la expresión de ROS1 y/o actividad quinasa ROS1 (por ejemplo, transcripción, traducción o estabilidad) se puede usar solo o en combinación con el compuesto de la Fórmula I, la, o compuesto 1 o una sal farmacéuticamente aceptable del mismo.
[0244] El agente inhibidor puede ser ROS1-específico o puede ser no específico (por ejemplo, inhibidores de quinasa no específicos, inhibidores multidiana). Se han desarrollado diferentes inhibidores de quinasa ROS1 y se han investigado sus aplicaciones clínicas. En dirección 3' de la actividad quinasa ROS1, existen quinasas, tales como fosfatidilinositol 3 quinasas (PI3K) y quinasas con señal extracelular 1/2 (ERK) (Wixted JH et al. J Biol Chem 2011), y STAT3 (Hwang JH et al. Mol Endocrinol 2003; 17: 1155-1166). Un fármaco, que inhibe tales rutas de transmisión de señal en dirección 3', también, se puede utilizar como una alternativa a un inhibidor de quinasa ROS1 o como un adyuvante solo o en combinación con los compuestos de la Fórmula 1.
[0245] En una realización de la presente divulgación, los sujetos, que se considera que tienen una translocación SLC34A2-ROS1 o CD74-FIG o una deleción FIG-ROS1, se considera, también, que tienen una mutación ROS1 en un sitio fuera del sitio de translocación. Un ejemplo de una mutación ROS1 es una mutación de activación, que permite actividad quinasa constitutiva. La mutación de activación ROS1 es una mutación arbitraria, que causa un aumento en la activación en comparación con la mutación de tipo salvaje. Por ejemplo, una mutación de activación ROS1 puede ocasionar activación ROS1 permanente. Puede incluso existir una variante secundaria de mutación de activación ROS1, que está asociado con o es debido al uso de un inhibidor ROS1. Las mutaciones, que provocan un aumento en actividad de señal ROS1, se producen debido a, por ejemplo, una mutación puntual de dominio quinasa, deleción, inserción, duplicación o inversión o combinación de dos o más de aquellos, que provocan un aumento en la señal ROS1. Para los sujetos, que se considera que tienen tanto una translocación de fusión ROS1 o la deleción, como se proporciona en el presente documento, como una mutación ROS1, se puede, también, tomar la decisión de tratarlos con un inhibidor de quinasa ROS1. Adicionalmente, se puede realizar el tratamiento/la terapia en ellos basándose en aquella decisión.
[0246] Como se describe anteriormente, la efectividad de tratamiento de cáncer utilizando un inhibidor de tirosina quinasa ROS1 se considera alta en un paciente, en el que se detecta un polinucleótido de fusión SLC34A2-ROS1 o CD74-ROS1 o FIG-ROS1 utilizando el procedimiento de la presente divulgación. Por esta razón, el tratamiento de cáncer se puede llevar a cabo de manera eficiente, cuando se administra un inhibidor de tirosina quinasa ROS1 de manera selectiva a aquellos pacientes de cáncer, quienes poseen el gen de fusión SLC34A2-ROS1, o CD74- ROS1, o FIG-ROS1 y el gen ROS1. Por consiguiente, la presente divulgación proporciona un procedimiento para tratar cáncer, que comprende una etapa de administración de un inhibidor de tirosina quinasa ROS1, que es la Fórmula I, Ia o compuesto 1 o una sal farmacéuticamente aceptable del mismo, a un paciente, en el que la efectividad de tratamiento de cáncer utilizando aquel inhibidor de tirosina quinasa ROS1 se determinó ser alta mediante el procedimiento de
diagnóstico mencionado anteriormente descrito en el presente documento.
[0247] En la presente divulgación, un "espécimen" no es solamente un espécimen biológico (por ejemplo, células, tejido, órgano, fluido (sangre, fluido linfático, etc.) fluido digestivo, esputo, fluido de lavado alveolar/bronquial, orina, deposición), pero, también, incluye extractos de ácido nucleico (extracto de ADN genómico, extracto de ARNm o preparación de ADNc o preparación de ARNc preparado a partir de extracto de ARNm) o extractos de proteínas obtenidas de estos especímenes biológicos. Este espécimen puede ser también uno, que se ha sometido a tratamiento con fijación de formalina, tratamiento con fijación de alcohol, tratamiento con congelación o tratamiento con incrustación en parafina.
[0248] Además, el ADN genómico, ARNm, ADNc o proteína se pueden preparar por un experto en la técnica después de seleccionar una técnica conocida adecuada considerando el tipo, el estado y demás del espécimen.
[0249] Adicionalmente a las sustancias mencionadas anteriormente (polinucleótidos, anticuerpos) como ingredientes activos, el fármaco de la presente divulgación puede contener otros ingredientes farmacéuticamente aceptables. Los ejemplos de tales ingredientes incluyen agentes tampón, emulsionantes, agentes de suspensión, estabilizadores, conservantes, solución salina fisiológica y demás. Como agentes tampón, se pueden utilizar fosfatos, citratos, acetatos y demás. Como emulsionantes se pueden utilizar goma arábiga, alginato de sodio, tragacanto y demás. Como agentes de suspensión se pueden utilizar monoestearato de glicerol, monoestearato de aluminio, metilcelulosa, carboximetilcelulosa, hidroximetilcelulosa, lauril sulfato de sodio y demás. Como estabilizadores se pueden utilizar propilenglicol, sulfito de dietilo, ácido ascórbico y demás. Como conservantes se pueden utilizar azida de sodio, cloruro de benzalconio, ácido paraoxibenzoico, clorobutanol y demás.
[0250] Además, adicionalmente a la preparación, que contiene polinucleótidos y anticuerpos, las preparaciones, tales como, un sustrato, un control positivo (por ejemplo, polipéptidos de fusión SLC34A2-ROS1, CD74-ROS1 o FIG-ROS1, polipéptidos de fusión SLC34A2-ROS1, CD74-ROS1 o FiG-ROS 1 o células, que los poseen, etc.) y un control negativo necesario para la detección del etiquetado agregado a polinucleótidos y anticuerpos, un reactivo contratinción (DAPI, etc.) utilizada en hibridación in situ o los similares, una molécula necesaria para detectar el anticuerpo (por ejemplo, un anticuerpo secundario, proteína G, proteína A) y solución tamponada utilizada durante la disolución o el lavado del anticuerpo se pueden combinar como un equipo para su uso en el procedimiento de la presente divulgación. El equipo puede incluir instrucciones para el uso del equipo. La presente divulgación proporciona, también, el equipo mencionado anteriormente para su uso en el procedimiento de la presente divulgación.
[0251] Los procedimientos de detección descritos en el presente documento son particularmente útiles, cuando se detecta una proteína de fusión, que consiste esencialmente en dominio N-terminal de un socio de fusión y dominio C-terminal de proteína ROS1, que codifica un dominio quinasa. La proteína de fusión puede ser proteína de fusión SLC34A2-ROS1, que consiste esencialmente en dominio N-terminal de proteína SLC34A2 o fragmento del mismo y dominio C-terminal de proteína ROS1, que codifica un dominio quinasa. La proteína de fusión puede ser proteína de fusión CD74-ROS 1, que consiste esencialmente en dominio N-terminal de proteína CD74 o un fragmento del mismo y dominio C-terminal de proteína ROS1, que codifica un dominio quinasa. La proteína de fusión puede ser proteína de fusión FIG-ROS1, que consiste esencialmente en dominio N-terminal de proteína FIG o un fragmento del mismo y dominio C-terminal de proteína ROS1, que codifica un dominio quinasa. Se puede utilizar el procedimiento para diagnosticar un cáncer de pulmón, particularmente cáncer de pulmón de células no pequeñas e incluye: detectar al menos una de una reorganización cromosómica que involucra ROS-1, o deleción en cromosoma 6; una proteína de fusión, donde la proteína se fusiona con otra proteína; un gen de fusión, que codifica la proteína de fusión; y la sobreexpresión de ROS1 comparada con una muestra estándar de un individuo sin un cáncer. Cuando una de las reorganizaciones cromosómicas descritas anteriormente seleccionada del grupo mencionado anteriormente se detecta en la muestra de prueba, el individuo para tratamiento de un inhibidor de ROS1, tal como, una Fórmula I, la o compuesto 1 o una sal farmacéuticamente aceptable del mismo.
Preparación del Compuesto 1
La preparación de N-(4-{[6,7-bis(metiloxi)quinolin-4-il]oxi}fenil)-N'-(4-fluorofenil)ciclopropano-1,1-dicarboxamida y la sal de (L)-malato del mismo.
[0252] La vía sintética utilizada en la preparación de N-(4-[6,7-bis(metiloxi)quinolin-4-il]oxi}fenil)-N'-(4-fluorofenil)ciclopropano-1,1-dicarboxamida y la sal de (L)-malato del mismo se representan en el Esquema 1:
Esquema 1
La Preparación de 4-Cloro-6,7-dimetoxi-quinolina
[0253] Se cargó de manera secuencial un reactivo con 6,7-dimetoxi-quinolina-4-ol (10,0 kg) y acetonitrilo (64,0 L). La mezcla resultante se calentó hasta aproximadamente 65 °C y se añadió oxicloruro de fósforo (POCh, 50,0 kg). Después de la adición de POCh, la temperatura de la mezcla de reacción se elevó hasta aproximadamente 80 °C. La reacción se consideró completada (aproximadamente 9.0 horas), cuando quedaron menos de 2 por ciento del material inicial (en proceso de análisis [HPLC] de cromatografía líquida de alto rendimiento). La mezcla de reacción se enfrió hasta aproximadamente 10 °C y, a continuación, se apagó en una solución fría de diclorometano (DCM, 238,0 kg), 30% de NH4OH (135,0 kg) y hielo (440,0 kg). La mezcla resultante se calentó hasta aproximadamente 14 °C y se separaron las fases. La fase orgánica se lavó con agua (40,0 kg) y se concentró utilizando destilación a vacío para eliminar el disolvente (aproximadamente 190,0 kg). Se añadió el éter metil-t butílico (MTBE, 50,0 kg) al lote y la mezcla se enfrió hasta aproximadamente 10 °C, tiempo durante el cual el producto se cristalizó. Se recuperaron los sólidos utilizando centrifugación, se lavaron con n-heptano (20,0 kg) y se secaron a aproximadamente 40 °C para proporcionar el compuesto principal (8,00).
La Preparación de 6,7-Dimetil-4-(4-nitro-fenoxi)-quinolina
[0254] Se cargó de manera secuencial un reactivo con 4-cloro-6,7-dimetoxi-quinolina (8,0 kg), 4 nitrofenol (7,0 kg), 4 dimetilaminopiridina (0,9 kg) y 2,6 lutidina (40,0 kg). Los contenidos de reactivo se calentaron hasta aproximadamente 147 °C. Cuando la reacción se finalizó (se quedaron menos de 5 por ciento de material inicial como se determinó en el proceso de análisis HPLC, aproximadamente 20 horas), los contenidos de reactivo se enfriaron hasta aproximadamente 25 °C. Se añadió metanol (26,0 kg), seguido de carbonato de potasio (3,0 kg) disuelto en agua (50,0 kg). Los contenidos de reactivo se agitaron durante aproximadamente 2 horas. La precipitación sólida resultante se filtró, se lavo con agua (67,0 kg) y se secó a 25 °C durante aproximadamente 12 horas para proporcionar el compuesto principal (4,0 kg).
La Preparación de 4-(6,7 -Dimetoxi-quinolina-4-iloxi)-fenilamina
[0255] Se añadió una solución, que contenía formiato de potasio (5,0 kg), ácido fórmico (3,0 kg) y agua (16,0 kg), a una mezcla de 6,7-dimetoxi-4-(4-nitro-fenoxi)-quinolina (4,0 kg), 10 por ciento de paladio sobre carbono (50 por ciento
humedecido con agua, 0,4 kg) en tetrahidrofurano (THF, 40,0 kg), que se calentó hasta aproximadamente 60 °C. La adición se llevó a cabo de tal modo, que la temperatura de la mezcla de reacción permaneció a aproximadamente 60 °C. Cuando la reacción se consideró completada como se determinó utilizando el análisis de HpLC en proceso (se quedaron menos de 2 por ciento de material inicial, típicamente 1,5 horas), los contenidos de reactivo se filtraron. El filtrado se concentró utilizando destilación a vacío a aproximadamente 35 °C hasta la mitad de su volumen inicial, que dio como resultado la precipitación del producto. El producto se recuperó mediante filtración, se lavó con agua (l2 ,0 kg) y se secó a vacío a aproximadamente 50 °C para proporcionar el compuesto principal (3,0 kg; 97 por ciento de área bajo curva (AUC)).
La Preparación de ácido 1-(4-Fluoro-fenilcarbamoil)-ciclopropanocarboxílico
[0256] Se añadió trietilamina (8,0 kg) a una solución enfriada (aproximadamente 4 °C) de ácido ciclopropano-1,1-dicarboxílico (2 1, 10,0 kg) disponible comercialmente en THF (63,0 kg) de tal forma que la temperatura del lote no superó 10 °C. La solución se agitó durante aproximadamente 30 minutos, y, a continuación, se añadió cloruro de tionilo (9,0 kg), manteniendo la temperatura de lote por debajo de 10 °C. Cuando la adición se completó, se añadió una solución de 4-fluoroanilina (9,0 kg) en THF (25,0 kg) de tal forma que la temperatura de lote no superó 10 °C. La mezcla se agitó durante aproximadamente 4 horas y, a continuación, se diluyó con acetato de isopropilo (87,0 kg). La solución se lavó secuencialmente con hidróxido de sodio acuoso (2,0 kg disueltos en 50,0 L de agua), agua (40,0 L) y cloruro de sodio (10,0 kg disueltos en 40,0 L de agua). La solución orgánica se concentró utilizando destilación al vacío seguida de la adición de heptano, que dio como resultado la precipitación de sólido. El sólido se recuperó utilizando centrifugación y, a continuación, se secó al vacío a aproximadamente 35 °C para proporcionar el compuesto principal. (10 ,0 kg).
La Preparación de cloruro de 1-(4-Fluoro-fenilcarbamoil)-ciclopropanocarbonilo
[0257] Se añadió cloruro de oxalilo (1,0 kg) a una solución de ácido 1-(4-fluoro-fenilcarbamoil)-ciclopropanocarboxílico (2,0 kg) en una mezcla de THF (11 kg) y N, N-dimetilformamida (DMF; 0,02 kg) de tal forma que la temperatura de lote no excedió 30 °C. La solución se utilizó en la siguiente etapa sin procesamiento adicional.
La preparación de N-(4-{[6,7-bis(metiloxi)quinolin-4-il]oxi}fenil)-N'-(4-fluorofenil)ciclopropano-1,1-dicarboxamida
[0258] Se añadió la solución de la etapa anterior, que contenía cloruro de 1-(4-fluoro-fenilcarbamoil)-ciclopropanocarbonilo a una mezcla de 4-(6,7-dimetoxi-quinolina-4-iloxi)-fenilamina (3,0 kg) y carbonato de potasio (4,0 kg) en THF (27,0 kg) y agua (13,0 kg) de tal modo que la temperatura de lote no excedió 30 °C. Cuando la reacción se completó (típicamente en 10 minutos), se añadió agua (74,0 kg). La mezcla se agitó a 15-30 °C durante aproximadamente 10 horas, que dio como resultado la precipitación del producto. El producto se recuperó mediante filtración, se lavó con una solución prefabricada de THF (11,0 kg) y agua (24,0 kg) y se secó al vacío a aproximadamente 64 °C durante aproximadamente 12 horas para proporcionar el compuesto principal (base libre, 5,0 kg). 1H NMR (400 MHz, da-DMSO): 510,2 (s, 1H), 10,05 (s, 1H), 8,4 (s, 1H), 7,8 (m, 2H), 7,65 (m, 2H), 7,5 (s, 1H), 7,35 (s, 1H), 7,25 (m, 2H), 7,15(m, 2H), 6,4 (s, 1H), 4,0 (d, 6 H), 1,5(s, 4H).
LC/MS: M+H= 502.
La preparación de N-(4-{[6,7-bis(metNoxi)qumoMn-4-N]oxi}fenM)-N'-(4-fluorofenM)ciclopropano-1,1-dicarboxamida, sal de (L)-malato
[0259] Se añadió una solución de ácido L-málico (2,0 kg) en agua (2,0 kg) a una solución de Ácido Ciclopropano-1,1-dicarboxílico con base libre de [4-(6,7-dimetoxi-quinolina-4-iloxi)-fenil]-amida (4-fluoro-fenil)-amida (1 5, 5,0 kg) en etanol, manteniendo una temperatura de lote de aproximadamente 25 °C. A continuación, se añadieron carbono (0,5 kg) y tiol sílice (0,1 kg) y la mezcla resultante se calentó hasta aproximadamente 78 °C, el punto en el que se añadió agua (6,0 kg). A continuación, la mezcla de reacción se filtró, seguido de la adición de isopropanol (38,0 kg) y se enfrió hasta aproximadamente 25 °C. El producto se recuperó mediante filtración y se lavó con isopropanol (20,0 kg) y se secó a aproximadamente 65 °C para proporcionar el compuesto principal (5,0 kg).
Preparación alternativa de N-(4-{[6,7-Bis(metiloxi)quinolin-4-il]oxi}fenil)-N '-(4-fluorofenil)ciclopropano-1,1-dicarboxamida y la sal de (L)-malato del mismo.
[0260] La vía sintética alternativa, que se puede utilizar para la preparación de N-(4{-[6,7-bis(metiloxi)quinolin-4-il]oxi}fenil)-N'-(4-fluorofenil)ciclopropano-1,1-dicarboxamida y la sal de (L)-malato del mismo se representan en el Esquema 2 como se describe en PCT/US2012/024591.
Esquema 2
Ácido (L)-málico
La Preparación de 4-Cloro-6,7-dimetoxi-quinolina
[0261] Se cargó de manera secuencial un reactor con 6,7-dimetoxi-quinolina-4-ol (47,0 kg) y acetonitrilo (318,8 L). La mezcla resultante se calentó hasta aproximadamente 60 °C y se añadió oxicloruro de fósforo (POCh, 130,6 kg). Después de la adición de POCh, la temperatura de la mezcla de reacción se elevó hasta aproximadamente 77 °C. La reacción se consideró completada (aproximadamente 13 horas), cuando quedaron menos de 3% del material inicial (análisis [HPLC] de cromatografía líquida de alto rendimiento en proceso). La mezcla de reacción se enfrió hasta aproximadamente 2-7 °C y, a continuación, se apagó en una solución fría de diclorometano (DCM, 482,8 kg), 26% de NH4OH (251,3 kg) y agua (900 L). La mezcla resultante se calentó hasta aproximadamente 20-25 °C y se separaron las fases. La fase orgánica se filtro a través de un lecho AW hiflo super-cel NF (Celite; 5,4 kg) y el lecho de filtro se lavó con DCM (118,9 kg). La fase orgánica combinada se lavó con salmuera (282,9 kg) y se mezcló con agua (120 L). Las fases se separaron y la fase orgánica se concentró utilizando destilación al vacío con la extracción de disolvente (aproximadamente 95 L de volumen residual). Se añadió DCM al reactivo, que contenía la fase orgánica y se concentró utilizando destilación al vacío con la extracción de disolvente (aproximadamente 90 L de volumen residual). A continuación, se añadió metil t-butil éter (MTBE, 226,0 kg) y la temperatura de la mezcla se ajusto hasta -20 a -25 °C y se mantuvo durante 2,5 horas, que dio como resultado precipitación de sólido, que se filtró a continuación y se lavó con n-heptano (92,0 kg) y se secó sobre un filtro a aproximadamente 25 °C bajo nitrógeno para proporcionar el compuesto principal. (35,6 kg).
La Preparación de 4-(6, 7-Dimetoxi-quinolina-4-iloxi)-fenilamina
[0262] 4-Aminofenol (24,4 kg) se disolvió en N,N-dimetilacetamida (DMA, 184,3 kg) se añadió a un reactivo, que contenía 4-cloro-6,7-dimetoxiquinolina (35,3 kg), t-butóxido de sodio (21,4 kg) y DMA (167,2 kg) a 20-25 °C. A continuación, esta mezcla se calentó hasta 100-105 °C durante aproximadamente 13 horas. Después de considerar la reacción completada como se determinó utilizando el análisis de HPLC en proceso (se quedaron menos de 2 por ciento de material inicial), los contenidos de reactivo se enfriaron a 15-20 °C y se añadió agua (preenfriada, 2-7 °C, 587 L) de tal modo para mantener la temperatura de 15-30 °C. El precipitado sólido resultante se filtró, se lavó con una mezcla de agua (47 L) y DMA (89,1 kg) y finalmente con agua (214 L). A continuación, la torta de filtración se secó a aproximadamente 25 °C sobre un filtro para producir 4-(6, 7-dimetoxi-quinolina-4-iloxi)-fenilamina bruta (59,4 kg húmedos, 41,6 kg secos calculados basándose en LOD). 4-(6, 7-dimetoxi-quinolina-4-iloxi)-fenilamina bruta se puso a reflujo (aproximadamente 75 °C) en una mezcla de tetrahidrofurano (THF, 211,4 kg) y DMA (108,8 kg) durante aproximadamente 1 hora y, a continuación, se enfrió hasta 0-5 °C y se envejeció durante 1 hora, después de que el sólido se filtró, se lavó con THF (147,6 kg) y se secó sobre un filtro al vacío a aproximadamente 25 °C para producir
4-(6,7-dimetoxi-quinolina-4-iloxi)-fenilamina (34,0 kg).
La Preparación Alternativa de 4-(6, 7-Dimetoxi-quinolina-4-iloxi)-fenilamina
[0263] Se añadieron 4-doro-6,7-dimetoxiquinolina (34,8 kg) y 4-aminofenol (30,8 kg) y tert pentóxido de sodio (1,8 equivalentes) 88,7 kg, 35 porcentaje en peso de THF) a reactivo, seguido de N,N-dimetilacetamida (DMA, 293,3 kg). A continuación, la mezcla se calentó hasta 105-105 °C durante aproximadamente 9 horas. Después de considerar la reacción completada como se determinó utilizando análisis de HPLC en proceso (se quedaron menos de 2 por ciento de material inicial), los contenidos de reactivo se enfriaron a 15-25 °C y se añadió agua durante el periodo de dos horas mientras se mantenía la temperatura entre 20-30 °C. A continuación, la mezcla de reacción se agitó durante una hora adicional a 20-25 °C. El producto bruto se recogió mediante la filtración y se lavó con una mezcla de 88 kg de agua y 82,1 kg de DMA seguido de 175 kg de agua. El producto se secó sobre un filtro de secado durante 53 horas. El LOD mostró menos de 1 por ciento de p/p.
[0264] En un procedimiento alternativo, se utilizaron 1,6 equivalentes de tert-pentóxido de sodio y la temperatura de reacción se aumentó desde 110-120 °C. Adicionalmente, la temperatura disminuida se aumentó a 35-40 °C y la temperatura inicial de la adición de agua se ajustó a 35-40 °C con un exotérmico permitido a 45 °C.
La Preparación de ácido 1-(4-Fluoro-fenilcarbamoil)-ciclopropanocarboxílico
[0265] Se añadió trietilamina (19,5 kg) a una solución enfriada (aproximadamente 5 °C) de ácido ciclopropano-1,1-dicarboxílico (24,7 kg) en THF (89,6 kg) de tal forma que la temperatura del lote no superó 5 °C. La solución se agitó durante aproximadamente 1,3 horas, y, a continuación, se añadió cloruro de tionilo (23,1 kg), manteniendo la temperatura de lote por debajo de 10 °C. Cuando la adición se completó, la solución se agitó durante aproximadamente 4 horas manteniendo la temperatura por debajo de 10 °C. A continuación, se añadió una solución de 4-fluoroanilina (18,0 kg) en THF (33,1 kg) de tal forma que la temperatura de lote no superó 10 °C. La mezcla se agitó durante aproximadamente 10 horas después de que la reacción se consideró completada. A continuación, la mezcla de reacción se diluyó con acetato de isopropilo (218,1 kg). Esta solución se lavó secuencialmente con hidróxido de sodio acuoso (10,4 kg, 50 por ciento disueltos en 119 L de agua), diluida más con agua (415 L), a continuación, con agua (100 L) y finalmente con cloruro de sodio acuoso (20,0 kg disueltos en 100 L de agua). La solución orgánica se concentró utilizando destilación al vacío (100 L de volumen residual) por debajo de 40 °C seguido de la adición de nheptano (171,4 kg), que dio como resultado la precipitación de sólido. El sólido se recuperó mediante filtración y se lavó con n-heptano (102,4 kg), que dio como resultado ácido 1-(4-fluoro-fenilcarbamoil)-ciclopropanocarboxílico (29,0 kg) en bruto. El ácido 1-(4-fluoro-fenilcarbamoil)-ciclopropanocarboxílico bruto se disolvió en metanol (139,7 kg) a aproximadamente 25 °C seguido de la adición de agua (320 L), que dio como resultado lechada, que se recuperó mediante filtración, se lavó secuencialmente con agua (20 L) y n-heptano (103,1 kg) y, a continuación, se secó sobre un filtro a aproximadamente 25 °C bajo nitrógeno para proporcionar el compuesto principal (25,4 kg).
La Preparación de cloruro de 1-(4-Fluoro-fenilcarbamoil)-ciclopropanocarbonilo
[0266] Se añadió cloruro de oxalilo (12,6 kg) a una solución de ácido 1-(4-fluoro-fenilcarbamoil)-ciclopropanocarboxílico (22,8 kg) en una mezcla de THF (96,1 kg) y N, N-dimetilformamida (DMF; 0,23 kg) de tal forma que la temperatura de lote no excedió 25 °C. La solución se utilizó en la siguiente etapa sin procesamiento adicional.
La Preparación de cloruro de 1-(4-Fluoro-fenilcarbamoil)-ciclopropanocarbonilo
[0267] Un reactivo se cargó con ácido 1-(4-fluoro-fenilcarbamoil)-ciclopropanocarboxílico (35 kg), 344 g de DMF, y 175 kg de THF. La mezcla de reacción se ajusto a 12-17 °C y, a continuación, a la mezcla de reacción se añadieron 19,9 kg de cloruro de oxalilo durante un periodo de 1 hora. La mezcla de reacción se agitó a 12-17 °C de 3 a 8 horas. La solución se utilizó en la siguiente etapa sin procesamiento adicional.
La preparación de [4-(6,7-dimetoxi-quinolina-4-iloxi)-fenil]-amida (4-fluoro-fenil)-amida de ácido ciclopropano-1,1-dicarboxílico
[0268] Se añadió la solución de la etapa anterior, que contenía cloruro de 1-(4-fluoro-fenilcarbamoil)-ciclopropanocarbonilo a una mezcla de compuesto de 4-(6,7-dimetoxi-quinolina-4-iloxi)-fenilamina (23,5 kg) y carbonato de potasio (31,9 kg) en THF (245,7 kg) y agua (116 L) de tal modo que la temperatura de lote no excedió 30 °C. Cuando la reacción se completó (en aproximadamente 20 minutos), se añadió agua (653 L). La mezcla se agitó a 20-25 °C durante aproximadamente 10 horas, que dio como resultado la precipitación del producto. El producto se recuperó mediante filtración, se lavó con una solución prefabricada de THF (68,6 kg) y agua (256 L) y se secó primero sobre un filtro bajo nitróge a aproximadamente 25 °C y, a continuación, a aproximadamente 45 °C al vacío para proporcionar el compuesto principal (41,0 kg, 38,1 kg, calculado basándose en LOD).
La Preparación Alternativa de [4-(6,7-dimetoxi-quinolina-4-iloxi)-fenil]-amida (4-fluoro-fenil)-amida de ácido ciclopropano-1,1-dicarboxílico
[0269] Un reactivo se cargó con 4-(6,7-dimetoxi-quinolina-4-iloxi)-fenilamina (35,7 kg, 1 equivalente), seguido de 412,9 kg de THF. A la mezcla de reacción se añadió una solución de 48,3 de K2CO3 en 169 kg de agua. La solución de cloruro de ácido descrita en la Preparación Alternativa de cloruro de 1-(4-Fluoro-fen¡lcarbamoil)-c¡clopropanocarbon¡lo anteriormente se transfirió al reactivo, que contenía 4-(6,7-dimetoxi-quinolina-4-iloxi)-fenilamina, mientras se mantenía la temperatura entre 20-30 °C durante un mínimo de dos horas. La mezcla de reacción se agitó a 20-25 °C durante un mínimo de tres horas. La temperatura de reacción se ajusto a continuación a 30-25 °C y la mezcla se agitó. La agitación se detuvo y las fases de la mezcla se separaron. La fase acuosa baja se removió y se descartó. A la fase orgánica superior se añadieron 804 kg de agua. La mezcla de reacción se agitó a 15-25 °C durante un mínimo de 16 horas.
[0270] El producto se precipitó. El producto se filtró y se lavó con una mezcla de 179 kg de agua y 157,9 kg de THF en dos partes. El producto bruto se secó al vacío durante un mínimo de dos horas. A continuación, el producto seco se absorbió en 285,1 kg de THF. La suspensión resultante se transfirió a un frasco de reacción y se agitó hasta que la suspensión se convirtió en una solución clara (disuelta), que requería calentamiento hasta 30-35 °C durante aproximadamente 30 minutos. A continuación, se añadieron 456 kg de agua a la solución, así como 20 kg de SDAG-1 etanol (etanol desnaturalizado con metanol durante dos horas). La mezcla se agitó a 15-25 °C durante al menos 16 horas. El producto se filtró y se lavó con una mezcla de 143 kg de agua y 126,7 de THF en dos partes. El producto se secó a una temperatura máxima de punto fijo de 40 °C.
[0271] En un procedimiento alternativo, la temperatura de reacción durante la formación de cloruro de ácido se ajustó a 10-15 °C. La temperatura de recristalización se cambió de 15-25 °C a 45-50 °C durante 1 hora y, a continuación, a 15-25 °C durante 2 horas.
La preparación de [4-(6,7-dimetoxi-quinolina-4-iloxi)-fenil]-amida (4-fluoro-fenil)-amida de ácido ciclopropano-1,1-dicarboxílico, sal de malato
[0272] Se añadieron [4-(6,7-dimetoxi-quinolina-4-iloxi)-fenil]-amida (4-fluoro-fenil)-amida de ácido ciclopropano-1,1-dicarboxílico (1-5; 13,3 kg), ácido L-málico (4,96 kg), metil etil cetona (MEK; 188,6 kg) y agua (37,3 kg) a un reactivo y la mezcla se calentó a reflujo (aproximadamente 74 °C) durante aproximadamente 2 horas. La temperatura de reactivo se redujo a 50-55 °C y los contenidos de reactivo se filtraron. Las etapas secuenciales descritas anteriormente se repitieron dos veces más empezando con cantidades similares de material inicial (13,3 kg), ácido L-málico (4,96 kg), MEK (198,6 kg) y agua (37,2 kg). El filtrado combinado se secó azeotrópicamente con presión atmosférica utilizando Me K (1133,2 kg) (volumen residual aproximado 711 L; KF < □ 0.5 % p/p) a aproximadamente 74 °C. La temperatura de los contenidos de reactivo se redujo hasta 20-25 °C y se mantuvo durante aproximadamente 4 horas, que dio como resultado precipitación de sólido, que se filtró, se lavó con MEK (448 kg) y se secó al vacío a 50 °C para proporcionar el compuesto principal (45,5 kg).
La Preparación Alternativa de [4-(6,7-dimetoxi-quinolina-4-iloxi)-fenil]-amida (4-fluoro-fenil)-amida de ácido ciclopropano-1,1-dicarboxílico, sal de (L) malato
[0273] Se añadieron [4-(6,7-dimetoxi-quinolina-4-iloxi)-fenil]-amida (4-fluoro-fenil)-amida de ácido ciclopropano-1,1-dicarboxílico (47,9 kg), ácido L-málico (17,2), 658,2 kg de metil etil cetona y 129,1 kg de agua (37,3 kg) a un reactivo y la mezcla se calentó a 50-55 °C durante aproximadamente 1-3 horas y, a continuación, a 55-60 °C durante 4-5 horas adicionales. La mezcla se aclaró mediante filtración a través de un cartucho de 1 pm. La temperatura de reactivo se ajustó a 20-25 °C y se destiló al vació a 150-200 mm HG con una temperatura máxima de camisa de 55 °C con el rango de volumen de 558-731 L.
[0274] La destilación al vacío se llevó a cabo dos veces más con la carga de 380 kg y 380,2 kg de metil etil cetona, respectivamente. Después de la tercera destilación, el volumen del lote se ajustó a 18 v/p de [4-(6,7-dimetoxi-quinolina-4-iloxi)-fenil]-amida (4-fluoro-fenil)-amida de ácido ciclopropano-1,1-carboxílico añadiendo 159,9 kg de metil etil cetona para proporcionar un volumen total de 880 L. Una destilación adicional al vacío se llevó a cabo ajustando 245,7 de metil etil cetona. La mezcla de reacción se agitó de manera moderada a 20-25 °C durante al menos 24 horas. El producto se filtró y se lavó con 415,1 kg de metil etil cetona en tres partes. El producto se secó al vacío con la temperatura de camisa en un punto fijo a 45 °C.
[0275] En un procedimiento alternativo, el orden de adición se cambió de tal modo, que una solución de 17,7 kg de ácido L-málico disuelto en 129,9 kg de agua se añadió a [4-(6,7-dimetoxi-quinolina-4-iloxi)-fenil]-amida (4-fluoro-fenil)-amida (48,7 kg) de ácido ciclopropano-1,1-dicarboxílico de en metil etil cetona (673,3 kg).
Ejemplo Biológico
Crecimiento de células HCC-78 y NCI-H2228
[0276] Se obtuvieron ambas células HCC-78 y NCI-H2228 de DSMZ y ATCC respectivamente como una muestra biológica congelada descongelada en un matraz volumétrico T-75 (Nunc) y crecidas hasta 80 o 90% de manera confluente para células adherentes o hasta que las células alcanzaron una densidad de 1 - 2 x 106 células/mL para las células de suspensión.
[0277] NCI-H2228, una línea celular de adenocarcinoma de NSCLC de pulmón humano (ATCC, CRL 5935), que alberga las fusiones, que activan EML4- ALK, se plantó con una densidad de 3,5 x 104 células/orificio y 2,5 x 104 células/orificio durante 48 horas o 72 horas respectivamente, en 0,1 ml en RPMI 1640 (ATCC 30-2001), 10 % de FBS, 1 % de penicilina-estreptomicina en placas de 96 orificios negros (Costar, 3904).
[0278] HCC-78, una línea celular de adenocarcinoma de NSCLC de pulmón humano (ATCC, ACC 563), que alberga las fusiones, que activan SLC34A2-ROS1 y fosfor-Met, se plantó con una densidad de 2,5 x 104 células/orificio y 2 x 104 células/orificio durante 48 horas o 72 horas respectivamente, en 0,1 ml en RPMI 1640 (ATCC 30-2001), 10 % de FBS (desactivado por calor), 1 % de penicilina-estreptomicina en placas de 96 orificios negros (Costar, 3904).
[0279] Las células crecieron a 37°C en una atmósfera de 5% de CO2 humedecida durante 16 horas. Se añadieron DMSO o disoluciones de serie del compuesto 1 en un medio, que contiene medio o suero libre de suero fresco, a células y se incubaron durante 48 horas o 72 horas a 37 °C en una atmósfera de 5% de CO2 humedecida antes de la adición de solución etiquetada con 5'-bromo-2'-deoxi-uridina (BrdU) (Roche) durante 2 a 4 horas. Antes de llevar a cabo la evaluación las células de suspensión se transfirieron a bandejas con filtro (Corning, 3504). Las células se evaluaron para la proliferación utilizando incorporación, que controla BrdU. Los reactivos de la Proliferación Celular de ELISA, equipo de BrdU (quimioluminiscencia) (Roche Biosciences, 11 669 915 001) se utilizaron según las instrucciones del fabricante. Después de etiquetar BrdU, las células se fijaron con solución FixDenat. Se añadió el conjugado de peroxidasa anti-BrdU (POD) y se lavaron las bandejas con 1x PBS. Se añadió la solución de sustrato y se realizaron las mediciones de luminiscencia hechas sobre un lector de placas Victor Wallac V (Perkin Elmer). El porcentaje de inhibición de proliferación se calculó comparando la proliferación celular durante el tratamiento con el compuesto 1 con muestras tratadas con DMSO. Los valores de porcentaje de inhibición de 5 a 8 de concentraciones del compuesto 1 se utilizaron para calcular los valores IC50.
Ejemplo Biológico
La inhibición de fosforilación de ROS1
[0280] Las células HCC-78, una línea celular de adenocarcinoma de NSCLC de pulmón humana (DSMZ, ACC 563), que alberga la fusión, que activa SLC34A2-ROS1, y fosfor-Met, se plantaron en placas de 6 orificios a 3 x105 células/orificio (Nunclon, 152795), RPMI 1640 (ATCC, 30-20019), que contiene 10% de FBS (desactivado por calor, Cellgro) y 1% de penicilina-estreptomicina (Cellgro). Se añadieron DMSO o disoluciones en serie del compuesto 1 en un medio fresco con 10% de FBS con una concentración final de 0,3% de DMSO (vehículo) a las células y se incubaron durante 3 horas. Después del tratamiento, las células se recogieron y se lavaron con PBS frío y se lisaron de inmediato con 0,15 mL de tampón de lisis frío (50 mM Tris HCl, pH 8.0, 150 mM NaCl, 1%de NP 40, 0,1% de SDS, 0,5% de deoxicolato de sodio, 1 mM de EDTA, 50 mM de NaF, 1 mM de pirofosfato de sodio, 1 mM de Na3VO4, 2 mM de PMSF, 10 |jg/mL de aprotinina,5 jg/mL de leupeptina y 5 jg/m L de pepstatina A). Se recogieron los lisados y se midieron las concentraciones de proteína utilizando el procedimiento de BCA (Pierce). Se recogieron los lisados y se midieron las concentraciones de proteína utilizando el procedimiento de BCA (Pierce). Se mezclaron los lisados con tampón de muestra NuPage LDS (Invitrogen NP0007) y Agente de Reducción (Invitrogen #NP0004), a continuación, se calentaron a 70 °C durante 20 minutos. Se cargaron 20 jg de proteína en NuPage con 10 % de geles Bis Tris (Invitrogen, NP0302). Las proteínas se transfirieron a membranas de celulosa (Invitrogen, LC2001), se bloquearon durante 1 hora en Tampón de Bloqueo Odyssey (Li-Cor,927 40000) y se incubaron a 4 °C durante la noche con el anticuerpo monoclonal de ratón anti-ROSI (1:1000) (Cell Signaling Technology, 3266) y anticuerpo de conejo Antifosfo ROS1 (Y2274) (1:1000) (Cell Signaling Technology, 3077) diluidos en Tampón de Bloqueo Odyssey, que contiene 0,1% de Tween 20. Las membranas se lavaron cuatro veces durante 10 minutos cada una con tampón TBS T (50 mM Tris HCl, pH7.2; 150 mM NaCl; 0,1% de Tween 20) y se secaron con anticuerpos secundarios de RDye680 de Cabra anti-Ratón (Li-Cor, 92632220) y IRDye800 Cabra anti-Conejo (Li-Cor, 92632211) en Tampón de Bloqueo Odyssey, que contenía 0,1% de Tween 20 y 0,01% de SDS durante 60 minutos a temperatura ambiente. Las membranas se lavaron cuatro veces durante 10 minutos cada una con tampón TBS-T y se enjuagaron con PBS dos veces. Las membranas se escanearon utilizando el Escaner Odyssey (Li-Cor) y se cuantifico la intensidad de señal de cada banda utilizando ImageQuant (Molecular Devices). Se calcularon los valores IC50 basándose en la señal con el tratamiento de compuesto en comparación con el control de vehículo (DMSO).
[0281] La inhibición de actividad quinasa mediada por SLC34A2-ROS1 en células de NSCLC HCC-78 administradas en dosis únicas escaladas del compuesto 1, o 3-[(1R)-1-(2,6-dicloro-3-fluorofenil)etoxi]-5-(1-piperidin-4-ilpirazol-4-il)piridin-2- amina (Crizotinib; XALKORI®) se muestra en la Figura 1. La Figura 1 muestra claramente la actividad inhibidora potente del compuesto 1 contra quinasa de fusión ROS1 y es más potente que el control de comparación, 3-[(1R)-1-(2,6-dicloro-3-fluorofenil)etoxi]-5-(1-piperidin-4-ilpirazol-4-il)piridin-2-amina (Crizotinib) con las concentraciones puestas a prueba in vitro.
La Proliferación Dependiente de ROS1 de Células NSCLC HCC-78
[0282] Los valores de IC50 se calcularon para el compuesto 1 y el control de comparador, 3-[(1R)-1-(2,6-dicloro-3-fluorofenil)etoxi]-5-(1-piperidin-4-ilpirazol-4-il)piridin-2-amina (Crizotinib) después de la incubación de agentes activos
Claims (15)
1. Compuesto para su uso en un procedimiento de tratamiento de adenocarcinoma de pulmón en un paciente, en el que el compuesto es el compuesto 1:
o una sal farmacéuticamente aceptable del mismo; y
en el que el adenocarcinoma de pulmón es cáncer de pulmón de células no pequeñas positivo en la fusión de SLC34A2-ROS1, CD74-ROS1 o FIG-ROS1.
2. Compuesto para su uso, según la reivindicación 1, en el que el compuesto es la sal de (L)-malato o (D)-malato del compuesto 1.
3. Compuesto para su uso, según la reivindicación 1 o la reivindicación 2, en el que el compuesto se administra como una composición farmacéutica que comprende adicionalmente un portador, excipiente o diluyente farmacéuticamente aceptable.
4. Compuesto para su uso, según cualquiera de las reivindicaciones anteriores, en el que el compuesto se administra después de tratamiento con cisplatino y/o gemcitabina; o en el que el compuesto se administra después de tratamiento con carboplatino; o en el que el compuesto se administra después de tratamiento con carboplatino y/o gemcitabina; o en el que el compuesto se administra después de tratamiento con cisplatino y/o carboplatino; o en el que el compuesto se administra después de tratamiento con docetaxel; o en el que el compuesto se administra después de tratamiento con crizotinib; o en el que el compuesto se administra después de tratamiento con crizotinib y/o gemcitabina; o en el que el compuesto se administra después de tratamiento con crizotinib y/o gemcitabina y/o docetaxel; o en el que el compuesto se administra después de tratamiento con cisplatino y/o gemcitabina y/o docetaxel.
7. Compuesto para su uso, según la reivindicación 6, en el que el cáncer mediado por quinasa ROS1 es adenocarcinoma de pulmón; más particularmente, en el que el adenocarcinoma de pulmón es cáncer de pulmón de células no pequeñas; más particularmente, en el que el adenocarcinoma de pulmón es cáncer de pulmón de células no pequeñas positivo en la fusión de SLC34A2-r Os 1, CD74-ROS1 o FIG-r Os 1.
8. Compuesto para su uso, según la reivindicación 6 o la reivindicación 7, en el que el compuesto se administra como una composición farmacéutica, que comprende el compuesto 1 o una sal farmacéuticamente aceptable del mismo y al menos un portador farmacéuticamente aceptable.
9. Compuesto para su uso, según la reivindicación 8, en el que la composición farmacéutica se administra diariamente durante más de 3 meses.
10. Compuesto para su uso, según la reivindicación 8 o la reivindicación 9, en el que la composición farmacéutica se administra en una dosis de 5, 10, 15, 20, 25, 30, 35, 40, 45, 50, 55, 65, 70, 75, 80, 85, 90 o 95 mg/día.
11. Compuesto para su uso, según cualquiera de las reivindicaciones 7-10, en el que la detección del cáncer de pulmón de células no pequeñas positivo en la fusión de SLC34A2-ROS1, CD74-ROS1 o FIG-ROS1 se realiza mediante un ensayo FISH, ClSH o SISH; o en el que la detección del cáncer de pulmón de células no pequeñas positivo en la fusión de SLC34A2-ROS1, CD74-r Os 1 o FIG-ROS1 se realiza utilizando cualquier forma de PCR genómico, secuenciación directa, secuenciación por PCR, ensayo RT-PCT o similares; o en el que la detección del cáncer de pulmón de células no pequeñas positivo en la fusión de SLC34A2-ROS1, CD74-ROS1 o FIG-ROS1 se realiza utilizando un anticuerpo que se une de forma específica con el polipéptido de fusión SLC34A2-ROS1, CD74-ROS1 o FIG-ROS1 o un fragmento del mismo.
12. Compuesto para su uso en un procedimiento de diagnóstico y tratamiento de un paciente, en el que el paciente tiene un tumor de NSCLC y el tumor se identifica como NSCLC positivo en la fusión de SLC34A2-ROS1, CD74-ROS1 o FIG-ROS1, en el que el compuesto es el compuesto 1:
o una sal farmacéuticamente aceptable del mismo, y en el que el compuesto se administra con al menos un portador farmacéuticamente aceptable.
13. Compuesto para su uso, según la reivindicación 12, en el que la identificación del cáncer de pulmón de células no pequeñas positivo en la fusión de SLC34A2-ROS1, CD74-ROS1 o FIG-ROS1 se realiza mediante un ensayo FISH, CISH o SISH; o en el que la identificación del cáncer de pulmón de células no pequeñas positivo en la fusión de SLC34A2-ROS1, CD74-ROS1 o FIG-ROS1 se realiza utilizando cualquier forma de PCR genómico, secuenciación directa, secuenciación por PCR, ensayo RT-PCT o similares; o en el que la identificación del cáncer de pulmón de células no pequeñas positivo en la fusión de SLC34A2-ROS1, CD74-ROS1 o FIG-ROS1 se realiza utilizando un anticuerpo, que se une de forma específica con polipéptido de fusión SLC34A2-ROS1, CD74-ROS1 o FIG-ROS1 o un fragmento del mismo.
14. Compuesto para su uso, según cualquiera de las reivindicaciones anteriores, en el que el compuesto produce al menos un efecto terapéutico seleccionado del grupo que consiste en la reducción de tamaño de un tumor, la reducción de la metástasis, remisión completa, remisión parcial, enfermedad estable, aumento de tasa global de respuesta y una respuesta completa patológica.
15. Compuesto para su uso, según cualquiera de las reivindicaciones anteriores, en el que el compuesto 1 se administra por vía oral una vez al día como su base libre o la sal de malato como una cápsula o comprimido, que contiene hasta 100 mg del compuesto 1, conteniendo preferiblemente 5, 10, 15, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 80, 85, 90, 95 o 100 mg del comprimido 1.
Applications Claiming Priority (2)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201461984599P | 2014-04-25 | 2014-04-25 | |
| PCT/US2015/027800 WO2015164869A1 (en) | 2014-04-25 | 2015-04-27 | Method of treating lung adenocarcinoma |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| ES2874875T3 true ES2874875T3 (es) | 2021-11-05 |
Family
ID=53055132
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| ES15721100T Active ES2874875T3 (es) | 2014-04-25 | 2015-04-27 | Procedimiento de tratamiento de adenocarcinoma de pulmón |
Country Status (21)
| Country | Link |
|---|---|
| US (1) | US20170042880A1 (es) |
| EP (2) | EP3134084B1 (es) |
| JP (2) | JP6696908B2 (es) |
| KR (1) | KR102474701B1 (es) |
| CN (1) | CN106488768A (es) |
| AR (1) | AR100191A1 (es) |
| AU (1) | AU2015249232B2 (es) |
| BR (1) | BR112016024672A2 (es) |
| CA (1) | CA2946416C (es) |
| DK (1) | DK3134084T3 (es) |
| EA (1) | EA035223B1 (es) |
| ES (1) | ES2874875T3 (es) |
| HU (1) | HUE054557T2 (es) |
| IL (1) | IL248408B (es) |
| MX (1) | MX375716B (es) |
| PL (1) | PL3134084T3 (es) |
| PT (1) | PT3134084T (es) |
| SG (1) | SG11201608657QA (es) |
| TW (1) | TWI724988B (es) |
| UA (1) | UA121655C2 (es) |
| WO (1) | WO2015164869A1 (es) |
Families Citing this family (15)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| JP2014513129A (ja) | 2011-05-02 | 2014-05-29 | エクセリクシス, インク. | 癌および骨癌疼痛の治療方法 |
| EA201691850A1 (ru) | 2014-03-17 | 2016-12-30 | Экселиксис, Инк. | Дозирование составов, содержащих кабозантиниб |
| MA40386A (fr) | 2014-07-31 | 2016-02-04 | Exelixis Inc | Procédé de préparation de cabozantinib marqué au fluor-18 et d'analogues de celui-ci |
| EA034992B1 (ru) | 2014-08-05 | 2020-04-15 | Экселиксис, Инк. | Комбинации лекарственных средств для лечения множественной миеломы |
| RU2748549C2 (ru) | 2016-04-15 | 2021-05-26 | Экселиксис, Инк. | Способ лечения почечно-клеточного рака с использованием n-(4-(6,7-диметоксихинолин-4-илокси)фенил)-n'-(4-фторфенил)циклопропан-1,1-дикарбоксамида, (2s)-гидроксибутандиоата |
| CN107353246A (zh) * | 2016-12-27 | 2017-11-17 | 辅仁药业集团熙德隆肿瘤药品有限公司 | 一种制备抗肿瘤药物卡博替尼的方法 |
| EP3631011B1 (en) | 2017-05-31 | 2021-06-23 | Roche Diagnostics GmbH | Multiplex pcr detection of alk, ret, and ros fusions |
| UA126402C2 (uk) | 2017-06-09 | 2022-09-28 | Екселіксіс, Інк. | Рідка лікарська форма для лікування раку |
| CN107541550A (zh) * | 2017-08-21 | 2018-01-05 | 上海派森诺生物科技股份有限公司 | 人ros1融合基因检测方法 |
| CN111492246A (zh) * | 2017-09-20 | 2020-08-04 | 梅尔莎纳医疗公司 | 用于预测对napi2b靶向疗法的响应的组合物和方法 |
| CN117624045A (zh) | 2018-01-26 | 2024-03-01 | 埃克塞里艾克西斯公司 | 用于治疗激酶依赖性病症的化合物 |
| WO2019148043A1 (en) | 2018-01-26 | 2019-08-01 | Exelixis, Inc. | Compounds for the treatment of kinase-dependent disorders |
| CR20250117A (es) | 2018-01-26 | 2025-05-09 | Exelixis Inc | COMPUESTOS PARA EL TRATAMIENTO DE TRASTORNOS DEPENDIENTES DE CINASAS (Divisional 2020-0355) |
| AR119069A1 (es) * | 2019-06-04 | 2021-11-24 | Exelixis Inc | Compuestos para el tratamiento de trastornos dependientes de quinasas |
| DE102020005002A1 (de) * | 2020-08-17 | 2022-02-17 | Epo Experimentelle Pharmakologie & Onkologie Berlin-Buch Gmbh | Mittel zur Therapie von Tumorerkrankungen |
Family Cites Families (14)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US4107288A (en) | 1974-09-18 | 1978-08-15 | Pharmaceutical Society Of Victoria | Injectable compositions, nanoparticles useful therein, and process of manufacturing same |
| US5447841A (en) | 1986-01-16 | 1995-09-05 | The Regents Of The Univ. Of California | Methods for chromosome-specific staining |
| US5756696A (en) | 1986-01-16 | 1998-05-26 | Regents Of The University Of California | Compositions for chromosome-specific staining |
| US5491224A (en) | 1990-09-20 | 1996-02-13 | Bittner; Michael L. | Direct label transaminated DNA probe compositions for chromosome identification and methods for their manufacture |
| US5145684A (en) | 1991-01-25 | 1992-09-08 | Sterling Drug Inc. | Surface modified drug nanoparticles |
| US6573043B1 (en) | 1998-10-07 | 2003-06-03 | Genentech, Inc. | Tissue analysis and kits therefor |
| CA2744997A1 (en) | 2003-09-26 | 2005-04-07 | Exelixis, Inc. | C-met modulators and method of use |
| US20100143918A1 (en) | 2007-01-19 | 2010-06-10 | Cell Signaling Technology, Inc. | Translocation and mutant ros kinase in human non-small cell lung carcinoma |
| WO2008083319A1 (en) | 2006-12-29 | 2008-07-10 | Il Yang Pharmaceutical Company, Ltd. | Solid state forms of enantiopure ilaprazole |
| EP3072963B1 (en) | 2007-10-18 | 2020-04-01 | Cell Signaling Technology, Inc. | Translocation and mutant ros kinase in human non-small cell lung carcinoma |
| KR101733773B1 (ko) * | 2009-01-16 | 2017-05-10 | 엑셀리시스, 인코포레이티드 | N-(4-{〔6,7-비스(메틸옥시)퀴놀린-4-일〕옥시}페닐)-n'-(4-플루오로페닐)사이클로프로판-1,1-디카르복사미드의 말산염 및 그 결정형 |
| DK2881402T3 (en) | 2009-02-12 | 2017-08-28 | Cell Signaling Technology Inc | Mutant ROS expression in human liver cancer |
| TWI585088B (zh) * | 2012-06-04 | 2017-06-01 | 第一三共股份有限公司 | 作爲激酶抑制劑之咪唑并[1,2-b]嗒衍生物 |
| CN104703600A (zh) * | 2012-09-07 | 2015-06-10 | 埃克塞里艾克西斯公司 | 用于治疗肺腺癌的met、vegfr和ret的抑制剂 |
-
2015
- 2015-04-27 EP EP15721100.4A patent/EP3134084B1/en not_active Revoked
- 2015-04-27 UA UAA201611881A patent/UA121655C2/uk unknown
- 2015-04-27 WO PCT/US2015/027800 patent/WO2015164869A1/en not_active Ceased
- 2015-04-27 MX MX2016013600A patent/MX375716B/es active IP Right Grant
- 2015-04-27 HU HUE15721100A patent/HUE054557T2/hu unknown
- 2015-04-27 US US15/305,854 patent/US20170042880A1/en not_active Abandoned
- 2015-04-27 TW TW104113427A patent/TWI724988B/zh active
- 2015-04-27 BR BR112016024672-1A patent/BR112016024672A2/pt not_active Application Discontinuation
- 2015-04-27 IL IL248408A patent/IL248408B/en unknown
- 2015-04-27 CA CA2946416A patent/CA2946416C/en active Active
- 2015-04-27 SG SG11201608657QA patent/SG11201608657QA/en unknown
- 2015-04-27 AR ARP150101255A patent/AR100191A1/es unknown
- 2015-04-27 ES ES15721100T patent/ES2874875T3/es active Active
- 2015-04-27 EA EA201692150A patent/EA035223B1/ru not_active IP Right Cessation
- 2015-04-27 DK DK15721100.4T patent/DK3134084T3/da active
- 2015-04-27 EP EP21162169.3A patent/EP3906921A1/en not_active Withdrawn
- 2015-04-27 CN CN201580022580.7A patent/CN106488768A/zh active Pending
- 2015-04-27 KR KR1020167033010A patent/KR102474701B1/ko active Active
- 2015-04-27 PT PT157211004T patent/PT3134084T/pt unknown
- 2015-04-27 AU AU2015249232A patent/AU2015249232B2/en active Active
- 2015-04-27 PL PL15721100T patent/PL3134084T3/pl unknown
- 2015-04-27 JP JP2016564236A patent/JP6696908B2/ja active Active
-
2019
- 2019-07-05 JP JP2019126230A patent/JP2019189646A/ja active Pending
Also Published As
| Publication number | Publication date |
|---|---|
| PT3134084T (pt) | 2021-05-11 |
| JP2017513908A (ja) | 2017-06-01 |
| AU2015249232B2 (en) | 2020-06-25 |
| CA2946416A1 (en) | 2015-10-29 |
| KR20160147934A (ko) | 2016-12-23 |
| SG11201608657QA (en) | 2016-11-29 |
| UA121655C2 (uk) | 2020-07-10 |
| MX375716B (es) | 2025-03-06 |
| CN106488768A (zh) | 2017-03-08 |
| IL248408B (en) | 2022-06-01 |
| TW201622723A (zh) | 2016-07-01 |
| CA2946416C (en) | 2022-07-19 |
| TWI724988B (zh) | 2021-04-21 |
| EP3906921A1 (en) | 2021-11-10 |
| JP6696908B2 (ja) | 2020-05-20 |
| EP3134084A1 (en) | 2017-03-01 |
| US20170042880A1 (en) | 2017-02-16 |
| DK3134084T3 (da) | 2021-05-03 |
| EA201692150A1 (ru) | 2017-02-28 |
| MX2016013600A (es) | 2017-04-27 |
| EP3134084B1 (en) | 2021-03-17 |
| KR102474701B1 (ko) | 2022-12-05 |
| WO2015164869A1 (en) | 2015-10-29 |
| EA035223B1 (ru) | 2020-05-18 |
| IL248408A0 (en) | 2016-11-30 |
| HUE054557T2 (hu) | 2021-09-28 |
| AR100191A1 (es) | 2016-09-14 |
| AU2015249232A1 (en) | 2016-11-10 |
| BR112016024672A2 (pt) | 2021-02-02 |
| PL3134084T3 (pl) | 2021-09-27 |
| JP2019189646A (ja) | 2019-10-31 |
| NZ725576A (en) | 2021-03-26 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| ES2874875T3 (es) | Procedimiento de tratamiento de adenocarcinoma de pulmón | |
| ES2726605T3 (es) | Inhibidores de MET, VEGFR y RET para usar en el tratamiento del adenocarcinoma de pulmón | |
| ES2995194T3 (en) | Method for the prognosis and treatment of cancer metastasis | |
| US20150057335A1 (en) | Novel fusion genes identified in lung cancer | |
| US20150177246A1 (en) | Fusion gene of cep55 gene and ret gene | |
| EP3190416B1 (en) | Biomarker for predicting sensitivity to protein kinase inhibitor and use thereof | |
| ES2681618T3 (es) | Marcadores asociados con inhibidores de WNT | |
| US20200010904A1 (en) | Method of predicting effects of cdc7 inhibitor | |
| HK40064013A (en) | Method of treating lung adenocarcinoma | |
| NZ725576B2 (en) | Method of treating lung adenocarcinoma | |
| HK1235005B (en) | Method of treating lung adenocarcinoma | |
| JP5119546B2 (ja) | 胃を原発巣とする消化管間質腫瘍の悪性化の診断法 | |
| HK40071406A (en) | Inhibitors of met, vegfr and ret for use in the treatment of lung adenocarcinoma | |
| US20120077734A1 (en) | Methods and assays for treating conditions in which macrophages play a pathogenic role | |
| WO2022229846A1 (en) | Treatment of cancer using a transforming growth factor beta receptor type 1 inhibitor | |
| HK1212248B (en) | Inhibitors of met, vegfr and ret for use in the treatment of lung adenocarcinoma |