ES2829295T3 - Detección de alto rendimiento de marcadores moleculares basados en AFLP y secuenciación de alto rendimiento - Google Patents
Detección de alto rendimiento de marcadores moleculares basados en AFLP y secuenciación de alto rendimiento Download PDFInfo
- Publication number
- ES2829295T3 ES2829295T3 ES17163116T ES17163116T ES2829295T3 ES 2829295 T3 ES2829295 T3 ES 2829295T3 ES 17163116 T ES17163116 T ES 17163116T ES 17163116 T ES17163116 T ES 17163116T ES 2829295 T3 ES2829295 T3 ES 2829295T3
- Authority
- ES
- Spain
- Prior art keywords
- restriction
- adapters
- sequence
- restriction fragments
- sample
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
- 238000012165 high-throughput sequencing Methods 0.000 title claims description 13
- 208000005652 acute fatty liver of pregnancy Diseases 0.000 title claims description 3
- 238000001514 detection method Methods 0.000 title description 23
- 239000012634 fragment Substances 0.000 claims abstract description 178
- 238000000034 method Methods 0.000 claims abstract description 53
- 108091008146 restriction endonucleases Proteins 0.000 claims abstract description 43
- 102000039446 nucleic acids Human genes 0.000 claims abstract description 28
- 108020004707 nucleic acids Proteins 0.000 claims abstract description 28
- 150000007523 nucleic acids Chemical class 0.000 claims abstract description 28
- 230000000295 complement effect Effects 0.000 claims abstract description 25
- 108091093088 Amplicon Proteins 0.000 claims abstract description 21
- 102000054765 polymorphisms of proteins Human genes 0.000 claims abstract description 11
- 239000002773 nucleotide Substances 0.000 claims description 54
- 125000003729 nucleotide group Chemical group 0.000 claims description 54
- 238000012163 sequencing technique Methods 0.000 claims description 49
- 230000003321 amplification Effects 0.000 claims description 32
- 238000003199 nucleic acid amplification method Methods 0.000 claims description 32
- 239000003550 marker Substances 0.000 claims description 21
- 230000002068 genetic effect Effects 0.000 claims description 9
- 238000013507 mapping Methods 0.000 claims description 6
- 238000003786 synthesis reaction Methods 0.000 claims description 4
- 238000004458 analytical method Methods 0.000 claims description 3
- 239000011324 bead Substances 0.000 claims description 3
- 230000015572 biosynthetic process Effects 0.000 claims description 3
- XPPKVPWEQAFLFU-UHFFFAOYSA-J diphosphate(4-) Chemical group [O-]P([O-])(=O)OP([O-])([O-])=O XPPKVPWEQAFLFU-UHFFFAOYSA-J 0.000 claims description 3
- 235000011180 diphosphates Nutrition 0.000 claims description 3
- 230000001804 emulsifying effect Effects 0.000 claims description 3
- 238000003205 genotyping method Methods 0.000 claims description 3
- 239000000839 emulsion Substances 0.000 claims description 2
- 238000011068 loading method Methods 0.000 claims description 2
- 230000002441 reversible effect Effects 0.000 claims description 2
- 239000007787 solid Substances 0.000 claims description 2
- 239000013615 primer Substances 0.000 description 90
- 108020004414 DNA Proteins 0.000 description 55
- 238000005516 engineering process Methods 0.000 description 32
- 238000003752 polymerase chain reaction Methods 0.000 description 18
- 102000053602 DNA Human genes 0.000 description 15
- 239000000499 gel Substances 0.000 description 11
- 102000004190 Enzymes Human genes 0.000 description 10
- 108090000790 Enzymes Proteins 0.000 description 10
- 108010042407 Endonucleases Proteins 0.000 description 9
- 102000004533 Endonucleases Human genes 0.000 description 9
- 230000037230 mobility Effects 0.000 description 9
- 108020004682 Single-Stranded DNA Proteins 0.000 description 8
- 238000001962 electrophoresis Methods 0.000 description 8
- 108091034117 Oligonucleotide Proteins 0.000 description 7
- 238000009396 hybridization Methods 0.000 description 6
- 239000000203 mixture Substances 0.000 description 6
- 238000002360 preparation method Methods 0.000 description 5
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 4
- 230000000875 corresponding effect Effects 0.000 description 4
- 230000029087 digestion Effects 0.000 description 4
- 238000001502 gel electrophoresis Methods 0.000 description 4
- 241000196324 Embryophyta Species 0.000 description 3
- 108091028043 Nucleic acid sequence Proteins 0.000 description 3
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 description 3
- 238000005251 capillar electrophoresis Methods 0.000 description 3
- 210000004027 cell Anatomy 0.000 description 3
- 239000005547 deoxyribonucleotide Substances 0.000 description 3
- 125000002637 deoxyribonucleotide group Chemical group 0.000 description 3
- 201000010099 disease Diseases 0.000 description 3
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 3
- 238000013537 high throughput screening Methods 0.000 description 3
- 239000002987 primer (paints) Substances 0.000 description 3
- 238000012545 processing Methods 0.000 description 3
- 108090000623 proteins and genes Proteins 0.000 description 3
- 238000012175 pyrosequencing Methods 0.000 description 3
- 238000005070 sampling Methods 0.000 description 3
- 239000007790 solid phase Substances 0.000 description 3
- 230000004544 DNA amplification Effects 0.000 description 2
- 241000282414 Homo sapiens Species 0.000 description 2
- 241001465754 Metazoa Species 0.000 description 2
- 229920005654 Sephadex Polymers 0.000 description 2
- 239000012507 Sephadex™ Substances 0.000 description 2
- ISAKRJDGNUQOIC-UHFFFAOYSA-N Uracil Chemical compound O=C1C=CNC(=O)N1 ISAKRJDGNUQOIC-UHFFFAOYSA-N 0.000 description 2
- 240000008042 Zea mays Species 0.000 description 2
- 235000002017 Zea mays subsp mays Nutrition 0.000 description 2
- 238000004873 anchoring Methods 0.000 description 2
- 230000008901 benefit Effects 0.000 description 2
- 238000006243 chemical reaction Methods 0.000 description 2
- 239000002299 complementary DNA Substances 0.000 description 2
- OPTASPLRGRRNAP-UHFFFAOYSA-N cytosine Chemical compound NC=1C=CNC(=O)N=1 OPTASPLRGRRNAP-UHFFFAOYSA-N 0.000 description 2
- 238000013461 design Methods 0.000 description 2
- 238000002474 experimental method Methods 0.000 description 2
- UYTPUPDQBNUYGX-UHFFFAOYSA-N guanine Chemical compound O=C1NC(N)=NC2=C1N=CN2 UYTPUPDQBNUYGX-UHFFFAOYSA-N 0.000 description 2
- 230000006872 improvement Effects 0.000 description 2
- 238000010348 incorporation Methods 0.000 description 2
- 230000013011 mating Effects 0.000 description 2
- 238000003976 plant breeding Methods 0.000 description 2
- 229920000642 polymer Polymers 0.000 description 2
- 238000011002 quantification Methods 0.000 description 2
- 238000010186 staining Methods 0.000 description 2
- 239000000126 substance Substances 0.000 description 2
- RWQNBRDOKXIBIV-UHFFFAOYSA-N thymine Chemical compound CC1=CNC(=O)NC1=O RWQNBRDOKXIBIV-UHFFFAOYSA-N 0.000 description 2
- GFFGJBXGBJISGV-UHFFFAOYSA-N Adenine Chemical compound NC1=NC=NC2=C1N=CN2 GFFGJBXGBJISGV-UHFFFAOYSA-N 0.000 description 1
- 229930024421 Adenine Natural products 0.000 description 1
- 108700028369 Alleles Proteins 0.000 description 1
- 208000024827 Alzheimer disease Diseases 0.000 description 1
- 208000002109 Argyria Diseases 0.000 description 1
- 108020004998 Chloroplast DNA Proteins 0.000 description 1
- 239000003155 DNA primer Substances 0.000 description 1
- 230000006820 DNA synthesis Effects 0.000 description 1
- 108010014303 DNA-directed DNA polymerase Proteins 0.000 description 1
- 102000016928 DNA-directed DNA polymerase Human genes 0.000 description 1
- 102000003960 Ligases Human genes 0.000 description 1
- 108090000364 Ligases Proteins 0.000 description 1
- 108020005196 Mitochondrial DNA Proteins 0.000 description 1
- 206010028980 Neoplasm Diseases 0.000 description 1
- 238000012408 PCR amplification Methods 0.000 description 1
- 239000012807 PCR reagent Substances 0.000 description 1
- 108091093037 Peptide nucleic acid Proteins 0.000 description 1
- 238000012356 Product development Methods 0.000 description 1
- CZPWVGJYEJSRLH-UHFFFAOYSA-N Pyrimidine Chemical compound C1=CN=CN=C1 CZPWVGJYEJSRLH-UHFFFAOYSA-N 0.000 description 1
- 108091028664 Ribonucleotide Proteins 0.000 description 1
- 101100439974 Streptococcus pneumoniae serotype 4 (strain ATCC BAA-334 / TIGR4) clpE gene Proteins 0.000 description 1
- 108010006785 Taq Polymerase Proteins 0.000 description 1
- 235000005824 Zea mays ssp. parviglumis Nutrition 0.000 description 1
- 235000016383 Zea mays subsp huehuetenangensis Nutrition 0.000 description 1
- 230000006978 adaptation Effects 0.000 description 1
- 229960000643 adenine Drugs 0.000 description 1
- 239000011543 agarose gel Substances 0.000 description 1
- 238000003975 animal breeding Methods 0.000 description 1
- 238000004364 calculation method Methods 0.000 description 1
- 201000011510 cancer Diseases 0.000 description 1
- 239000003153 chemical reaction reagent Substances 0.000 description 1
- 238000003776 cleavage reaction Methods 0.000 description 1
- 235000005822 corn Nutrition 0.000 description 1
- 230000002596 correlated effect Effects 0.000 description 1
- 238000004163 cytometry Methods 0.000 description 1
- 229940104302 cytosine Drugs 0.000 description 1
- 238000007405 data analysis Methods 0.000 description 1
- 238000012217 deletion Methods 0.000 description 1
- 230000037430 deletion Effects 0.000 description 1
- 238000004925 denaturation Methods 0.000 description 1
- 230000036425 denaturation Effects 0.000 description 1
- 230000008021 deposition Effects 0.000 description 1
- 238000003745 diagnosis Methods 0.000 description 1
- 238000007720 emulsion polymerization reaction Methods 0.000 description 1
- 230000009144 enzymatic modification Effects 0.000 description 1
- 238000006911 enzymatic reaction Methods 0.000 description 1
- ZMMJGEGLRURXTF-UHFFFAOYSA-N ethidium bromide Chemical compound [Br-].C12=CC(N)=CC=C2C2=CC=C(N)C=C2[N+](CC)=C1C1=CC=CC=C1 ZMMJGEGLRURXTF-UHFFFAOYSA-N 0.000 description 1
- 229960005542 ethidium bromide Drugs 0.000 description 1
- 235000013305 food Nutrition 0.000 description 1
- 238000005194 fractionation Methods 0.000 description 1
- 238000013467 fragmentation Methods 0.000 description 1
- 238000006062 fragmentation reaction Methods 0.000 description 1
- 102000054766 genetic haplotypes Human genes 0.000 description 1
- 238000013090 high-throughput technology Methods 0.000 description 1
- 238000000338 in vitro Methods 0.000 description 1
- 238000003780 insertion Methods 0.000 description 1
- 230000037431 insertion Effects 0.000 description 1
- 235000009973 maize Nutrition 0.000 description 1
- 230000002438 mitochondrial effect Effects 0.000 description 1
- 235000016709 nutrition Nutrition 0.000 description 1
- 230000035764 nutrition Effects 0.000 description 1
- 239000000825 pharmaceutical preparation Substances 0.000 description 1
- 229940127557 pharmaceutical product Drugs 0.000 description 1
- 125000002924 primary amino group Chemical group [H]N([H])* 0.000 description 1
- 238000000746 purification Methods 0.000 description 1
- 230000002285 radioactive effect Effects 0.000 description 1
- 238000009790 rate-determining step (RDS) Methods 0.000 description 1
- 230000009467 reduction Effects 0.000 description 1
- 238000011160 research Methods 0.000 description 1
- 238000012552 review Methods 0.000 description 1
- 239000002336 ribonucleotide Substances 0.000 description 1
- 125000002652 ribonucleotide group Chemical group 0.000 description 1
- 230000007017 scission Effects 0.000 description 1
- 238000002864 sequence alignment Methods 0.000 description 1
- 241000894007 species Species 0.000 description 1
- 230000005477 standard model Effects 0.000 description 1
- 238000010561 standard procedure Methods 0.000 description 1
- 238000006467 substitution reaction Methods 0.000 description 1
- 230000008685 targeting Effects 0.000 description 1
- 238000012360 testing method Methods 0.000 description 1
- 229940113082 thymine Drugs 0.000 description 1
- 210000001519 tissue Anatomy 0.000 description 1
- 229940035893 uracil Drugs 0.000 description 1
- 230000004304 visual acuity Effects 0.000 description 1
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6844—Nucleic acid amplification reactions
- C12Q1/6853—Nucleic acid amplification reactions using modified primers or templates
- C12Q1/6855—Ligating adaptors
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6844—Nucleic acid amplification reactions
- C12Q1/6853—Nucleic acid amplification reactions using modified primers or templates
Landscapes
- Chemical & Material Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Organic Chemistry (AREA)
- Engineering & Computer Science (AREA)
- Zoology (AREA)
- Wood Science & Technology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Health & Medical Sciences (AREA)
- Biophysics (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Immunology (AREA)
- Microbiology (AREA)
- Molecular Biology (AREA)
- Analytical Chemistry (AREA)
- Physics & Mathematics (AREA)
- Biotechnology (AREA)
- Biochemistry (AREA)
- Bioinformatics & Cheminformatics (AREA)
- General Engineering & Computer Science (AREA)
- General Health & Medical Sciences (AREA)
- Genetics & Genomics (AREA)
- Measuring Or Testing Involving Enzymes Or Micro-Organisms (AREA)
Abstract
Metodo para detectar polimorfismos en fragmentos de restriccion de dos o mas muestras, que comprende los pasos de: (a) proporcionar dos o mas muestras de acido nucleico; (b) digerir cada muestra de acido nucleico con al menos una endonucleasa de restriccion para obtener un conjunto de fragmentos de restriccion; (c) proporcionar adaptadores sinteticos bicatenarios que comprenden - una secuencia compatible con el cebador 5', seguida de una seccion identificadora especifica de muestra, y - un extremo 3' que se puede ligar al extremo romo o saliente de un fragmento de restriccion; (d) ligar los adaptadores sinteticos bicatenarios a los fragmentos de restriccion en el conjunto, para proporcionar un conjunto de fragmentos de restriccion ligados a adaptadores; (e) opcionalmente amplificar el conjunto de fragmentos de restriccion ligados a adaptadores, con uno o mas cebadores que son al menos complementarios a: - la seccion identificadora especifica de muestra, - una seccion que es complementaria a los restos de la secuencia de reconocimiento de la endonucleasa de restriccion, para proporcionar fragmentos de restriccion ligados a adaptadores amplificados (amplicones); (f) determinar la secuencia de al menos - la seccion identificadora especifica de muestra; - los restos de la secuencia de reconocimiento de la endonucleasa de restriccion; y, - parte de la secuencia del fragmento de restriccion situada adyacente a los restos de la secuencia de reconocimiento de la endonucleasa de restriccion; y (g) detectar polimorfismos en la secuencia de los fragmentos de restriccion ligados a adaptadores en las dos o mas muestras obtenidas en el paso (f).
Description
DESCRIPCIÓN
Detección de alto rendimiento de marcadores moleculares basados en AFLP y secuenciación de alto rendimiento
Campo de la invención
[0001] La presente invención se refiere al campo de la biología molecular y la biotecnología. En particular, la invención se refiere al campo de la identificación y la detección de ácidos nucleicos. Más en particular la invención se refiere a métodos para la detección y la identificación de marcadores, en particular marcadores moleculares. La invención se refiere a la provisión de métodos de alto rendimiento para la detección y la identificación de marcadores moleculares. La invención se refiere además a la aplicación del método en la identificación y/o la detección de secuencias de nucleótidos que están relacionadas con una amplia variedad de rasgos genéticos, genes, haplotipos y combinaciones de los mismos. La invención se puede usar en el campo de la detección de alto rendimiento y la identificación de marcadores moleculares de cualquier origen, sea vegetal, animal, humano, artificial u otro.
Antecedentes de la invención
[0002] La comunidad científica, médica en particular, ha deseado durante mucho tiempo la exploración del ADN genómico. El ADN genómico es la clave para la identificación, el diagnóstico y el tratamiento de las enfermedades tales como el cáncer y la enfermedad de Alzheimer. Además de la identificación y el tratamiento de las enfermedades, la exploración del ADN genómico puede proporcionar ventajas significativas a los esfuerzos en mejora vegetal y animal, que pueden proporcionar respuestas a los problemas de alimentación y nutrición en el mundo.
[0003] Muchas enfermedades son conocidas por estar asociadas a componentes genéticos específicos, en particular a polimorfismos en genes específicos. La identificación de polimorfismos en muestras grandes tales como los genomas es en la actualidad una tarea laboriosa y que conlleva mucho tiempo. Sin embargo, dicha identificación es de gran valor para áreas tales como la investigación biomédica, el desarrollo de productos farmacéuticos, la tipificación de tejidos, la genotipificación y los estudios poblacionales.
[0004] Los marcadores, es decir, los marcadores genéticos, han sido usados durante mucho tiempo como un método de tipificación genética, es decir, para conectar un rasgo fenotípico con la presencia, la ausencia o la cantidad de una parte particular del ADN (gen). Una de las tecnologías de tipificación genética más versátil es la de AFLP, ya existente durante muchos años y ampliamente aplicable a cualquier organismo (para revisiones véanse Savelkoul et al. J. Clin. Microbiol, 1999, 37 (10), 3083-3091; Bensch et al. Molecular Ecology, 2005, 14, 2899-2914).
[0005] La tecnología de AFLP (Zabeau & Vos, 1993; Vos et al., 1995) ha encontrado un uso amplio en la mejora vegetal y otros campos desde su invención a principios de la década de los noventa. Esto se debe a varias características de AFLP, de las cuales la más importante es que no se necesita ninguna información de secuencia previa para generar un gran número de marcadores genéticos r\ de una forma reproducible. Además, el principio de amplificación selectiva, un principio básico de AFLP, asegura que el número de fragmentos amplificados se puede ajustar a la resolución del sistema de detección, sin tener en cuenta el origen o el tamaño del genoma.
[0006] La detección de fragmentos AFLP se realiza comúnmente por electroforesis en geles planos (Vos et al., 1995) o electroforesis capilar (van der Meulen et al., 2002). La mayoría de marcadores de AFLP clasificados de esta manera representan polimorfismos (de nucleótido único) que ocurren o en los sitios de reconocimiento de enzimas de restricción usadas para la preparación del modelo de AFLP o en sus nucleótidos flanqueantes cubiertos por cebadores de AFLP selectivos. El resto de los marcadores de AFLP son polimorfismos de inserción/deleción que ocurren en las secuencias internas de los fragmentos de restricción y una fracción muy pequeña en sustituciones de nucleótido único que ocurren en fragmentos de restricción pequeños (< aproximadamente 100 pb), que, para estos fragmentos, causan variaciones de movilidad reproducibles entre ambos alelos que se pueden observar en la electroforesis; estos marcadores de AFLP se pueden clasificar codominantemente sin tener que depender de las intensidades de banda.
[0007] Por lo tanto, en una huella de AFLP típica, los marcadores de AFLP constituyen la minoría de fragmentos amplificados (menos de un 50 por ciento, pero a menudo menos de un 20 por ciento), mientras que el resto se denominan comúnmente fragmentos AFLP constantes. Sin embargo, estos últimos son útiles en el procedimiento de clasificación en gel, ya que sirven como puntos de anclaje para calcular movilidades de fragmentos de los marcadores de AFLP y ayudar en la cuantificación de los marcadores para la clasificación codominante. La clasificación codominante (clasificación según la homocigosidad o la heterocigosidad) de los marcadores de AFLP
está actualmente restringida al contexto de obtención de huellas de una población segregante. En un panel de líneas no relacionadas, solo es posible la clasificación dominante.
[0008] Aunque el rendimiento de AFLP es muy alto debido a altos niveles de multiplexado en los pasos de amplificación y detección, el paso limitante de la velocidad es la potencia de resolución de la electroforesis. La electroforesis permite la identificación única de la mayoría de fragmentos amplificados basándose en la combinación de combinaciones de enzimas de restricción (EC), combinaciones de cebadores (PC) y movilidad, pero la electroforesis solo es capaz de distinguir los fragmentos amplificados en función de las diferencias de movilidad. Los fragmentos de movilidad similar se encuentran a menudo como las denominadas 'bandas apiladas' y, con la electroforesis, no se le puede prestar ninguna atención a la información que está contenida en las denominadas 'bandas constantes', es decir, fragmentos de restricción amplificados que no parecen diferir entre las especies comparadas. Además, en un sistema basado en gel típico, o en un sistema capilar tal como un MegaBACE, las muestras deben correrse en paralelo y solo pueden analizarse aproximadamente 100-150 bandas por carril en un gel o por capilar. Estas limitaciones obstaculizan también el rendimiento.
[0009] Idealmente, el sistema de detección debería ser capaz de determinar toda la secuencia de los fragmentos amplificados para capturar todos los fragmentos de restricción amplificados. Sin embargo, la mayoría de tecnologías de secuenciación de alto rendimiento no pueden proporcionar aún lecturas de secuenciación que abarquen fragmentos AFLP enteros, que tienen típicamente 100-500 pb de longitud.
[0010] Hasta ahora, la detección de marcadores/secuencias AFLP por secuenciación no ha sido económicamente realizable debido a, entre otras limitaciones, las limitaciones de los costes de la tecnología de secuenciación didesoxi de Sanger y otras tecnologías de secuenciación convencionales.
[0011] La detección por secuenciación en vez de la determinación de la movilidad aumentará el rendimiento porque:
1) Los polimorfismos situados en las secuencias internas se detectarán en la mayoría de (o en todos) los fragmentos amplificados; esto aumentará el número de marcadores por PC considerablemente.
2) No habrá ninguna pérdida de marcadores de AFLP debida a la comigración de marcadores de AFLP y bandas constantes.
3) La clasificación codominante no depende de la cuantificación de las intensidades de bandas y es independiente de la relación de los individuos de los que se han obtenido las huellas.
[0012] Sin embargo, la detección por secuenciación de todo el fragmento de restricción sigue siendo relativamente no económica. Además, el estado de la técnica actual de la tecnología de secuenciación tal como se describe aquí en otra parte (de 454 Life Sciences, www.454.com y Solexa, www.solexa.com), a pesar de su potencia de secuenciación sobrecogedora, solo puede proporcionar fragmentos de secuenciación de longitud limitada. Además, los métodos actuales no permiten el procesamiento simultáneo de muchas muestras en una ronda.
Definiciones
[0013] En la descripción y los ejemplos siguientes se utilizan una serie de términos. Para proporcionar una comprensión clara y consistente de la especificación y las reivindicaciones, incluido el alcance que ha de darse a tales términos, se proporcionan las definiciones siguientes. A menos que se defina de otro modo aquí, todos los términos técnicos y científicos usados tienen el mismo significado que entiende comúnmente una persona experta en la materia a la que pertenece esta invención.
[0014] Ácido nucleico: un ácido nucleico conforme a la presente invención puede incluir cualquier polímero u oligómero de las bases pirimidínicas y púricas, preferiblemente citosina, timina y uracilo, y adenina y guanina, respectivamente (véase Albert L. Lehniriger, Principles of Biochemistry, en 793-800 (Worth Pub. 1982)). La presente invención contempla cualquier desoxirribonucleótido, ribonucleótido o componente de ácido nucleico peptídico, y cualquier variante química de los mismos, tales como las formas metiladas; hidroximetiladas o glicosiladas de estas bases, y similares. Los polímeros u oligómeros pueden tener una composición heterogénea u homogénea, y se pueden aislar de fuentes de origen natural o se pueden producir artificial o sintéticamente. Además, los ácidos nucleicos pueden ser de ADN o de ARN, o una mezcla de los mismos, y pueden existir de manera permanente o transitoria en la forma monocatenaria o bicatenaria, incluidos homodúplex, heterodúplex y estados híbridos.
[0015] AFLP: AFLP se refiere a un método para la amplificación selectiva de ácidos nucleicos que se basa en la digestión de un ácido nucleico con una o más endonucleasas de restricción para producir fragmentos de restricción, el ligamiento de adaptadores a los fragmentos de restricción y la amplificación de los fragmentos de restricción
ligados a los adaptadores con al menos un cebador que sea complementario (en parte) al adaptador, complementario (en parte) a los restos de la endonucleasa de restricción, y que además contenga al menos un nucleótido seleccionado de forma aleatoria de entre A, C, T o G (o U, según el caso). AFLP no requiere ninguna información de secuencia previa y se puede realizar en cualquier ADN de partida. En general, AFLp comprende los pasos de:
(a) digerir un ácido nucleico, en particular un ADN o ADNc, con una o más endonucleasas de restricción específicas, para fragmentar el ADN en una serie correspondiente de fragmentos de restricción;
(b) ligar los fragmentos de restricción así obtenidos con un adaptador oligonucleotídico sintético bicatenario, un extremo del cual es compatible con uno o los dos extremos de los fragmentos de restricción, para producir así, fragmentos de restricción ligados a adaptadores, preferiblemente etiquetados, del ADN de partida; (c) poner en contacto los fragmentos de restricción ligados a adaptadores, preferiblemente etiquetados, bajo condiciones de hibridación con uno o más cebadores oligonucleotídicos que contienen nucleótidos selectivos en sus extremos 3';
(d) amplificar el fragmento de restricción ligado a un adaptador, preferiblemente etiquetado, hibridado con los cebadores por PCR o una técnica similar para causar un alargamiento adicional de los cebadores hibridados a lo largo de los fragmentos de restricción del ADN de partida con los que los cebadores han hibridado; y (e) detectar, identificar o recuperar el fragmento de ADN amplificado o alargado así obtenido.
[0016] AFLP proporciona así un subconjunto reproducible de fragmentos ligados a adaptadores. AFLP se describe en la EP 534858, la US 6045994 y en Vos et al. Se hace referencia a estas publicaciones para detalles adicionales con respecto a AFLP. El AFLP se usa comúnmente como una técnica de reducción de la complejidad y una tecnología de obtención de huellas genéticas. En el contexto del uso de AFLP como una tecnología de obtención de huellas, se ha desarrollado el concepto de un marcador de AFLP.
[0017] Marcador de AFLP: un marcador de AFLP es un fragmento de restricción ligado a un adaptador amplificado que es diferente entre dos muestras que han sido amplificadas usando AFLP (se han obtenido sus huellas), usando el mismo conjunto de cebadores. Como tal, la presencia o la ausencia de este fragmento de restricción ligado a un adaptador amplificado se puede usar como un marcador que está enlazado a un rasgo o fenotipo. En la tecnología de gel convencional, un marcador de AFLP aparece como una banda en el gel localizada con una determinada movilidad. En otras técnicas electroforéticas como la electroforesis capilar puede que no se haga referencia a esto como una banda, pero el concepto es el mismo, es decir, un ácido nucleico con una longitud y una movilidad determinadas. La ausencia o la presencia de la banda puede ser indicativa de (o estar asociada a) la presencia o la ausencia del fenotipo. Los marcadores de AFLP implican típicamente SNPs en el sitio de restricción de la endonucleasa o los nucleótidos selectivos. Ocasionalmente, los marcadores de AFLP pueden implicar indels en el fragmento de restricción.
[0018] Banda constante: una banda constante en la tecnología de AFLP es un fragmento de restricción ligado a un adaptador amplificado que es relativamente invariable entre muestras. Así, una banda constante en la tecnología de AFLP aparecerá, en un rango de muestras, en aproximadamente la misma posición en el gel, es decir, tiene la misma longitud/movilidad. En AFLP convencional estas se usan típicamente para anclar los carriles correspondientes a las muestras en un gel o los electroferogramas de múltiples muestras AFLP detectadas por electroforesis capilar. Típicamente, una banda constante es menos informativa que un marcador de AFLP. Sin embargo, como los marcadores de AFLP habituales implican SNPs en los nucleótidos selectivos o el sitio de restricción, las bandas constantes pueden comprender SNPs en los propios fragmentos de restricción, convirtiendo a las bandas constantes en una fuente alternativa interesante de información genética que es complementaria a los marcadores de AFLP.
[0019] Base selectiva: 'situada en el extremo 3' del cebador que contiene una parte que es complementaria al adaptador y una parte que es complementaria a los restos del sitio de restricción, la base selectiva se selecciona de forma aleatoria de entre A, C, T o G. Extendiendo un cebador con una base selectiva, la amplificación posterior producirá solo un subconjunto reproducible de los fragmentos de restricción ligados a adaptadores, es decir, solo los fragmentos que se pueden amplificar usando el cebador que lleva la base selectiva. Se pueden añadir nucleótidos selectivos al extremo 3' del cebador en un número variable entre 1 y 10. Típicamente 1-4 bastan. Ambos cebadores pueden contener un número variable de bases selectivas. Con cada base selectiva añadida, el subconjunto reduce la cantidad de fragmentos de restricción ligados a adaptadores amplificados en el subconjunto por un factor de aproximadamente 4. Típicamente, el número de bases selectivas usadas en AFLP se indica por N+M, donde un cebador lleva N nucleótidos selectivos y los otros cebadores llevan M nucleótidos selectivos. Así, un AFLP Eco/Mse 1/+2 es la forma abreviada para la digestión del ADN de partida con EcoRI y MseI, el ligamiento de adaptadores apropiados y la amplificación con un cebador dirigido a la posición restringida por EcoRI que lleva una base selectiva y el otro cebador dirigido al sitio restringido por MseI que lleva 2 nucleótidos selectivos. Un cebador usado en AFLP que lleva al menos un nucleótido selectivo en su extremo 3' también se representa como un AFLP-cebador. Los cebadores que no llevan un nucleótido selectivo en su extremo 3' y que son, de hecho, complementarios al adaptador y los restos del sitio de restricción se indican a veces como cebadores de AFLP+0.
[0020] Agolpamiento: el término "agolpamiento" se refiere a la comparación de dos o más secuencias de nucleótidos en función de la presencia de extensiones cortas o largas de nucleótidos idénticos o similares. Se conocen en la técnica varios métodos para el alineamiento de secuencias de nucleótidos, como se explicará adicionalmente a continuación. A veces, los términos "ensamblaje" o "alineamiento" se usan como sinónimos.
[0021] Identificador: una secuencia corta que se puede añadir a un adaptador o un cebador o incluir en su secuencia o, en su defecto, usarse como un marcador para proporcionar un identificador único. Tal identificador de secuencia puede ser una secuencia de bases única de longitud variable pero definida usada únicamente para identificar una muestra de ácido nucleico específica. Por ejemplo, las etiquetas de 4 pb permiten 4(exp4) = 256 etiquetas diferentes. Ejemplos típicos son las secuencias ZIP, conocidas en la técnica como etiquetas comúnmente usadas para la detección única por hibridación (Iannone et al. Cytometry 39:131-140, 2000). Usando dicho identificador, se puede determinar el origen de una muestra de PCR tras un procesamiento adicional. En caso de combinar productos procesados que se originan a partir de muestras de ácido nucleico diferentes, las muestras de ácido nucleico diferentes se identifican generalmente usando diferentes identificadores.
[0022] Secuenciación: el término secuenciación se refiere a la determinación del orden de los nucleótidos (secuencias de bases) en una muestra de ácido nucleico, por ejemplo ADN o ARN.
[0023] Cribado de alto rendimiento: el cribado de alto rendimiento, a menudo abreviado como HTS, es un método para la experimentación científica especialmente pertinente para los campos de la biología y la química. A través de una combinación de robótica moderna y otro hardware de laboratorio especializado, permite que un investigador cribe eficazmente grandes cantidades de muestras simultáneamente.
[0024] Endonucleasa de restricción: una endonucleasa de restricción o enzima de restricción es una enzima que reconoce una secuencia de nucleótidos específica (sitio diana) en una molécula de ADN bicatenario, y escindirá ambas cadenas de la molécula de ADN en o cerca de cada sitio diana.
[0025] Fragmentos de restricción: las moléculas de ADN producidas por digestión con una endonucleasa de restricción se denominan fragmentos de restricción. Cualquier genoma dado (o ácido nucleico, independientemente de su origen) será digerido por una endonucleasa de restricción particular en un conjunto discreto de fragmentos de restricción. Los fragmentos de ADN que se originan a partir de la escisión por endonucleasas de restricción se pueden usar adicionalmente en una variedad de técnicas y pueden, por ejemplo, ser detectados por electroforesis en gel.
[0026] Electroforesis en gel: para detectar fragmentos de restricción, puede requerirse un método analítico para fraccionar moléculas de ADN basándose en el tamaño. La técnica más frecuentemente usada para conseguir dicho fraccionamiento es la electroforesis en gel (capilar). La velocidad a la que se mueven los fragmentos de ADN en dichos geles depende de su peso molecular; así, las distancias recorridas se reducen a medida que las longitudes de los fragmentos aumentan. Los fragmentos de ADN fraccionados por electroforesis en gel se pueden visualizar directamente por un procedimiento de tinción, por ejemplo tinción de plata o tinción usando bromuro de etidio, si el número de fragmentos incluidos en el patrón es suficientemente pequeño. Alternativamente el tratamiento adicional de los fragmentos de ADN puede incorporar marcadores detectables en los fragmentos, tales como fluoróforos o marcadores radiactivos, que se usan preferiblemente para marcar una cadena del producto AFLP.
[0027] Ligamiento: la reacción enzimática catalizada por una enzima ligasa en la que dos moléculas de ADN bicatenario se unen de manera covalente se denomina ligamiento. En general, ambas cadenas de ADN se unen de manera covalente, pero también es posible evitar el ligamiento de una de las dos cadenas a través de la modificación química o enzimática de uno de los extremos de las cadenas. En ese caso la unión covalente ocurrirá en solo una de las dos cadenas de ADN.
[0028] Oligonucleótido sintético: las moléculas de ADN monocatenario con preferiblemente de aproximadamente 10 a aproximadamente 50 bases que se pueden sintetizar químicamente se denominan oligonucleótidos sintéticos. En general, estas moléculas de ADN sintéticas están diseñadas para tener una secuencia de nucleótidos única o deseada, aunque es posible sintetizar familias de moléculas que tienen secuencias relacionadas y que tienen diferentes composiciones de nucleótidos en posiciones específicas de la secuencia de nucleótidos. El término oligonucleótido sintético se usará para referirse a moléculas de ADN con una secuencia diseñada o deseada de nucleótidos.
[0029] Adaptadores: moléculas de ADN bicatenario cortas con un número limitado de pares de bases, por ejemplo de aproximadamente 10 a aproximadamente 30 pares de bases de longitud, que están diseñadas de manera que se puedan ligar a los extremos de los fragmentos de restricción. Los adaptadores están generalmente compuestos por dos oligonucleótidos sintéticos que tienen secuencias de nucleótidos que son parcialmente complementarias entre sí. Cuando se mezclan los dos oligonucleótidos sintéticos en solución bajo condiciones apropiadas, se
aparearán entre sí formando una estructura bicatenaria. Después del apareamiento, un extremo de la molécula de adaptador está diseñada de modo que sea compatible con el extremo de un fragmento de restricción y se puede ligar al mismo; el otro extremo del adaptador se puede diseñar de modo que no pueda ligarse, pero no es necesario que sea así (adaptadores ligados doblemente).
[0030] Fragmentos de restricción ligados a adaptadores: fragmentos de restricción que han sido cubiertos por adaptadores.
[0031] Cebadores: en general, el término cebadores se refiere a cadenas de ADN que pueden promover la síntesis de ADN. La ADN-polimerasa no puede sintetizar ADN de novo sin cebadores: solo puede extender una cadena de ADN existente en una reacción en la que la cadena complementaria se usa como un modelo para dirigir el orden de los nucleótidos que se van a ensamblar. Nos referiremos a las moléculas de oligonucleótido sintéticas que se usan en una reacción en cadena de la polimerasa (PCR) como cebadores.
[0032] Amplificación de ADN: el término amplificación de ADN se usará típicamente para indicar la síntesis in vitro de moléculas de ADN bicatenario usando una PCR. Cabe señalar que existen otros métodos de amplificación y que se pueden usar en la presente invención sin apartarse de lo esencial.
Resumen de la invención
[0033] La presente invención se define en las reivindicaciones anexas.
[0034] Los presentes inventores han descubierto que los problemas descritos anteriormente y otros problemas en la técnica se pueden superar concibiendo una manera genérica donde la versatilidad y la aplicabilidad de la tecnología de marcadores (AFLP) se pueda combinar con la de la tecnología de secuenciación de alto rendimiento del estado de la técnica.
[0035] Así, los presentes inventores han descubierto que la incorporación de un identificador específico de muestra en el fragmento de restricción ligado a un adaptador y/o la determinación de solo parte de la secuencia del fragmento de restricción proporciona una mejora muy eficiente y fiable de las tecnologías existentes. Se ha observado que, mediante la incorporación de un identificador específico de muestra, se pueden secuenciar múltiples muestras en una única ronda y, mediante la secuenciación de solo parte del fragmento de restricción, se puede conseguir una identificación adecuada del fragmento de restricción.
Breve descripción de los dibujos
[0036]
Figura 1: es una representación esquemática de la estructura del adaptador que se usa en un método basado en AFLP habitual para la secuenciación de etiquetas cortas para la detección de AFLP. Se muestra un fragmento AFLP típico derivado de un producto de digestión de una muestra de ADN con EcoRI y MseI y un ligamiento del adaptador posterior, seguido de un adaptador típico para el sitio EcoRI. El adaptador comprende, del extremo 5' al 3', una secuencia de cebador 5', que es opcional, y se puede usar para anclar cebadores de amplificación o para anclar el fragmento ligado a un adaptador a una esfera o superficie. Además, se muestra un identificador (dado como NNNNNN en una forma degenerada), seguido de restos de una secuencia de reconocimiento de una enzima de restricción (en esta EcoRI, es decir, AATTC). El último nucleótido del identificador preferiblemente no comprende una G para destruir el sitio de restricción EcoRI. Se proporciona un cebador adecuado que comprende la secuencia de cebador 5' opcional, un ejemplo de un cebador específico (ACTGAC), restos del sitio de reconocimiento y una sección que puede contener uno o más nucleótidos selectivos en el extremo 3'.
Figura 2: es una representación esquemática de la forma de realización donde se incorpora una secuencia de reconocimiento para una endonucleasa de restricción de tipo IIs en el adaptador. Después de la restricción con la enzima de tipo IIs, se pueden ligar adaptadores compatibles de tipo IIs a uno o los dos fragmentos restringidos A y B. El adaptador de tipo IIs comprende una secuencia de unión (o anclaje) de cebadores opcional, un identificador y una sección que contiene nucleótidos (degenerados) (NN) para hibridar con la proyección del sitio de restricción IIs. El cebador asociado puede contener uno o más nucleótidos selectivos (XYZ) en su extremo 3'.
Descripción detallada de la invención
[0037] En un aspecto, la presente descripción se refiere a un método para la identificación de fragmentos de restricción en una muestra, que comprende los pasos de:
(a) proporcionar un ácido nucleico de muestra;
(b) digerir el ácido nucleico de muestra con al menos una endonucleasa de restricción para obtener un conjunto de fragmentos de restricción;
(c) proporcionar adaptadores sintéticos bicatenarios que comprenden
- una secuencia compatible con el cebador 5',
- una sección identificadora específica de muestra,
- una sección que sea complementaria a los restos de la secuencia de reconocimiento de la endonucleasa de restricción;
(d) ligar los adaptadores sintéticos bicatenarios a los fragmentos de restricción en el conjunto, para proporcionar un conjunto de fragmentos de restricción ligados a adaptadores;
(e) amplificar el conjunto de fragmentos de restricción ligados a adaptadores, con uno o más cebadores que son al menos complementarios a:
- la sección identificadora específica de muestra,
- la sección que es complementaria a los restos de la secuencia de reconocimiento de la endonucleasa de restricción, para proporcionar fragmentos de restricción ligados a adaptadores amplificados (amplicones);
(f) determinar la secuencia de al menos la sección identificadora específica de muestra, los restos de la secuencia de reconocimiento de la endonucleasa de restricción y de parte de la secuencia del fragmento de restricción situada adyacente a la misma de (parte de) los fragmentos de restricción ligados a adaptadores amplificados.
(g) identificar la presencia o la ausencia de fragmentos de restricción ligados a adaptadores amplificados en la muestra.
[0038] Tratando un ácido nucleico de muestra de esta manera, se obtiene un conjunto de fragmentos de restricción amplificados por cada muestra que se secuencie. Cada fragmento de restricción se puede identificar como originado a partir de cierta muestra a través del identificador específico de muestra que es diferente para cada muestra. La secuenciación de los fragmentos de restricción ligados a adaptadores amplificados proporciona información de secuencia en al menos parte del fragmento de restricción ligado a un adaptador. La información contenida en la parte derivada del adaptador contiene información acerca de la muestra de la que se obtiene el fragmento, mientras que la información de secuencia del fragmento de restricción mismo proporciona información acerca del fragmento de restricción y permite la identificación del fragmento de restricción. Esta información de secuencia en el fragmento de restricción se utiliza para identificar el fragmento de restricción con una exactitud que depende del número de nucleótidos que se determine y el número de fragmentos de restricción en el conjunto de fragmentos de restricción ligados a adaptadores amplificados.
[0039] Para proporcionar una solución al problema de variación de muestreo que afecta a la exactitud de la identificación de marcadores moleculares por secuenciación contenidos en un conjunto de múltiples fragmentos, los presentes inventores también han descubierto que la detección de marcadores a través de la secuenciación se realiza preferiblemente con suficiente redundancia (profundidad) para muestrear todos los fragmentos amplificados al menos una vez y acompañada por medios estadísticos que abordan el problema de la variación de muestreo en relación a la exactitud de los genotipos llamados. Además, al igual que con la clasificación AFLP, en el contexto de una población segregante, la clasificación simultánea de los individuos parentales en un experimento, ayudará a determinar el umbral estadístico.
[0040] Así, en determinadas formas de realización, la redundancia de los fragmentos de restricción ligados a adaptadores amplificados y etiquetados es al menos 6, preferiblemente al menos 7, más preferiblemente al menos 8 y más preferiblemente al menos 9. En determinadas formas de realización, la secuencia de cada fragmento de restricción ligado a un adaptador se determina al menos 6, preferiblemente al menos 7, más preferiblemente al menos 8 y más preferiblemente al menos 9 veces. En determinadas formas de realización, la redundancia se selecciona de modo que, suponiendo una probabilidad general de 50/50 de identificar el locus correctamente como homocigótico, la probabilidad de identificar correctamente el locus sea más de un 95%, 96%, 97%, 98%, 99%, 99,5%.
[0041] A este respecto puede ser ilustrativo el cálculo siguiente: la tecnología de secuenciación de Solexa como se describe en este caso en otra parte, proporciona 40.000.000 de lecturas de aproximadamente 25 pb cada una, alcanzando el asombroso total de 1 mil millones pb en una única ronda. Suponiendo una redundancia en el muestreo de 10 veces, se pueden evaluar 4.000.000 de fragmentos únicos en una ronda. La combinación de 100 muestras permite secuenciar 40.000 fragmentos por cada muestra. Visto desde la perspectiva de AFLP, esto equivale a 160 combinaciones de cebadores con 250 fragmentos cada uno.
[0042] Este método permite la identificación de fragmentos de restricción de manera que es diferente de la detección de marcadores convencional a base de electroforesis.
[0043] En el primer paso del método para la identificación de fragmentos de restricción se proporciona un ácido nucleico de muestra. Los ácidos nucleicos en la muestra estarán usualmente en forma de A d N . Sin embargo, la información de secuencia de nucleótidos contenida en la muestra puede ser de cualquier fuente de ácidos nucleicos, incluidos, por ejemplo, ARN, ARN poliA+, ADNc, ADN genómico, ADN organular tal como ADN mitocondrial o de cloroplastos, ácidos nucleicos sintéticos, genotecas de ADN (tal como genotecas de BAC/clones de BAC agrupados), bancos de clones o cualquier selección o combinación de los mismos. El ADN en la muestra de ácido nucleico puede ser bicatenario, monocatenario y ADN bicatenario desnaturalizado en ADN monocatenario. La muestra de ADN puede ser de cualquier organismo, ya sea vegetal, animal, sintético o humano.
[0044] La muestra de ácido nucleico se restringe (o digiere) con al menos una endonucleasa de restricción para proporcionar un conjunto de fragmentos de restricción. En determinadas formas de realización, se pueden usar dos o más endonucleasas para obtener fragmentos de restricción. La endonucleasa puede ser un cortador frecuente (una secuencia de reconocimiento de 3-5 pb, tal como MseI) o un cortador raro (secuencia de reconocimiento de >5 pb, tal como EcoRI). En determinadas formas de realización preferidas, se prefiere una combinación de un cortador raro y uno frecuente. En determinadas formas de realización, en particular cuando la muestra contiene o deriva de un genoma relativamente grande, se puede preferir usar una tercera enzima (cortador raro o frecuente) para obtener un conjunto mayor de fragmentos de restricción de tamaño más corto.
[0045] Como endonucleasas de restricción, cualquier endonucleasa bastará. Típicamente, se prefieren endonucleasas de tipo II tales como EcoRI, MseI, PstI, etc. En determinadas formas de realización, se puede usar una endonucleasa de tipo IIs, es decir, una endonucleasa cuya secuencia de reconocimiento está localizada distante del sitio de restricción, es decir, tales como AceIII, AlwI, AlwXI, Alw26I, BbvI, BbvII, BbsI, BccI, Bce83I, BcefI, BcgI, BinI, BsaI, BsgI, BsmAI, BsmFl, BspMI, EarI, EciI, Eco31I, Eco57I, Esp3I, FauI, FokI, GsuI, HgaI, HinGUII, HphI, Ksp632I, MboII, MmeI, MnlI, NgoVIII, PleI, RleAI, SapI, SfaNI, TaqJI y Zthll lII. El uso de este tipo de endonucleasa de restricción lleva a ciertas adaptaciones al método, como se describirá aquí en otra parte.
[0046] Los fragmentos de restricción pueden tener extremos romos o extremos salientes, dependiendo de la endonucleasa usada. A estos extremos, pueden ligarse adaptadores. Típicamente, los adaptadores usados en la presente invención tienen un diseño particular. Los adaptadores usados en la presente invención pueden comprender una secuencia compatible con el cebador 5', que puede ser opcional para proporcionar una longitud suficiente del adaptador para el posterior apareamiento del cebador, seguido de una sección identificadora específica de muestra que puede comprender de 4 a 16 nucleótidos. Preferiblemente el identificador específico de muestra no contiene 2 o más bases idénticas consecutivas para evitar ultralecturas durante el paso de secuenciación. Además, en el caso de que se combinen 2 o más muestras y se utilicen múltiples identificadores específicos de muestra para distinguir el origen de las muestras, hay preferiblemente una diferencia entre los identificadores específicos de muestra de al menos 2, preferiblemente 3 pb. Esto permite una discriminación mejorada entre los diferentes identificadores específicos de muestra dentro de un grupo combinado de muestras. En el extremo 3' del adaptador se localiza una sección que es complementaria a los restos de la secuencia de reconocimiento de la endonucleasa de restricción. Por ejemplo, EcoRI reconoce 5-GAATTC-3' y corta entre G y AATTC. Por lo tanto, para EcoRI, la sección complementaria a los restos de la secuencia de reconocimiento de la endonucleasa de restricción es un nucleótido C.
[0047] El adaptador se liga (se conecta de manera covalente) con uno o ambos lados del fragmento de restricción. Cuando se realiza una digestión con más de una endonucleasa, se pueden usar diferentes adaptadores que darán lugar a diferentes conjuntos de fragmentos de restricción ligados a adaptadores.
[0048] Los fragmentos de restricción ligados a adaptadores se amplifican posteriormente con un conjunto de uno o más cebadores. El cebador puede ser complementario solo al adaptador, es decir, amplificación no selectiva. El cebador contiene preferiblemente una sección que es complementaria al identificador específico de muestra y una sección que es complementaria a los restos de la secuencia de reconocimiento de la endonucleasa de restricción. En determinadas formas de realización, el cebador puede contener en su extremo 3' uno o más nucleótidos selectivos para proporcionar un subconjunto de fragmentos de restricción ligados a adaptadores amplificados. El cebador puede, en su extremo 5', contener también nucleótidos adicionales para ayudar al anclaje del cebador a los fragmentos de restricción ligados a adaptadores. En determinadas formas de realización, el cebador puede contener nucleótidos que expresan características de hibridación mejoradas tales como LNAs o PNAs. Para amplificar fragmentos de restricción ligados a adaptadores de muestras combinadas en un grupo es posible usar conjuntos de cebadores degenerados, es decir, conjuntos de cebadores donde, para cada muestra, el identificador de muestra correspondiente se incorpora en el cebador. En determinadas formas de realización, es posible usar conjuntos de cebadores donde la sección identificadora es completamente degenerada (o al menos en gran parte), es decir, (casi) cada combinación de nucleótidos se proporciona en la sección identificadora específica de muestra. En combinación con condiciones de hibridación rigurosas en la amplificación y el uso opcional de nucleótidos de
tipo LNA o PNA para aumentar las características de hibridación, esto puede conducir a una amplificación muy eficiente.
[0049] La amplificación de los fragmentos de restricción ligados a adaptadores llevan a un conjunto de fragmentos de restricción ligados a adaptadores amplificados, denominados a veces amplicones.
[0050] Los amplicones (o al menos parte de los mismos) se someten a un paso que comprende al menos la determinación de la secuencia del identificador específico de muestra para determinar el origen del fragmento y de parte de la secuencia del fragmento de restricción. En la práctica esto equivale también a la determinación de las secciones situadas entremedias tales como los restos de la secuencia de reconocimiento de la endonucleasa de restricción. Secuenciando el identificador específico de muestra en combinación con parte del fragmento situado adyacente a la secuencia derivada del adaptador, es posible identificar únicamente fragmentos de restricción. Cuando se correlaciona con la presencia o la ausencia de un fenotipo, estos fragmentos de restricción identificados únicamente se pueden usar como marcadores moleculares. Esto permite la definición de una nueva generación de marcadores y equivale por lo tanto a una tecnología de marcadores novedosa con la versatilidad probada de la tecnología de AFLP, pero que es adecuada para las tecnologías de alto rendimiento y es generalmente aplicable entre cualquier tipo de organismo o ácido nucleico. La identificación de manera única de fragmentos de restricción en una muestra por determinación de parte de su secuencia mediante este método se puede repetir para múltiples muestras. La presencia o la ausencia de los fragmentos de restricción con la secuencia representada en la muestra es indicativa de la presencia o la ausencia de un fenotipo.
[0051] Una ventaja adicional de la tecnología de marcadores actualmente inventada basada en la combinación de AFLP y la secuenciación de alto rendimiento es la información adicional que se puede obtener en comparación con la tecnología de AFLP convencional. En AFLP, los amplicones que se designan como marcadores de AFLP contienen típicamente un polimorfismo en el sitio de reconocimiento, el sitio de restricción u, opcionalmente, en los nucleótidos selectivos. Los polimorfismos situados más alejados en el fragmento de restricción típicamente no se califican como marcadores de AFLP (aparte de quizás los polimorfismos de indels). Con el presente paso de secuenciación, también se determinan los nucleótidos adyacentes a los nucleótidos selectivos opcionales y esto lleva a la identificación de un mayor número de marcadores moleculares y a una mejora en la tecnología de marcadores existente.
[0052] La secuenciación de alto rendimiento usada en la presente invención es un método de experimentación científica especialmente pertinente para los campos de la biología y la química. A través de una combinación de robótica moderna y otro hardware de laboratorio especializado, permite que un investigador cribe eficazmente grandes cantidades de muestras simultáneamente.
Se prefiere que la secuenciación se realice usando métodos de secuenciación de alto rendimiento, tales como los métodos descritos en la WO 03/004690, la WO 03/054142, la WO 2004/069849, la WO 2004/070005, la WO 2004/070007 y la WO 2005/003375 (todas en nombre de 454 Life Sciences), por Seo et al. (2004) Proc. Natl. Acad. Sci. USA 101:5488-93, y las tecnologías de Helios, Solexa, US Genomics, etc.
Tecnología de 454 Life Sciences
[0053] En determinadas formas de realización, se prefiere que la secuenciación se realice usando el aparato y/o el método descrito en la WO 03/004690, la WO 03/054142, la WO 2004/069849, la WO 2004/070005, la WO 2004/070007 y la WO 2005/003375 (todas en nombre de 454 Life Sciences). La tecnología descrita permite la secuenciación de 40 millones de bases en una única ronda y es 100 veces más rápida y menos costosa que la tecnología competidora. La tecnología de secuenciación consiste en aproximadamente 5 pasos: 1) fragmentación del ADN y ligamiento de adaptadores específicos para crear una genoteca de ADN monocatenario (ssADN); 2) apareamiento de ssADN a esferas, emulsión de las esferas en microrreactores de agua en aceite y realización de PCR en emulsión para amplificar las moléculas de ssADN individuales en esferas; 3) selección de enriquecimiento para las esferas que contienen moléculas de ssADN amplificado en su superficie, 4) deposición de esferas que llevan ADN en una placa PicoTiter™; y 5) secuenciación simultánea en 100.000 pocillos por generación de una señal luminosa de pirofosfato. El método se explicará con más detalle a continuación.
En una forma de realización preferida, la secuenciación comprende los pasos de:
(a) aparear fragmentos adaptados a esferas, donde cada esfera se aparea con un único fragmento adaptado; (b) emulsionar las esferas en microrreactores de agua en aceite, donde cada microrreactor de agua en aceite comprende una única esfera;
(c) cargando las esferas en pocillos, donde cada pocillo comprende una única esfera; y generar una señal de pirofosfato.
En el primer paso (a), los adaptadores de secuenciación se ligan a fragmentos de la genoteca de combinación. Dicho adaptador de secuenciación incluye al menos una región "clave" para aparearse a una esfera, una región
de cebador de secuenciación y una región de cebador de PCR. Así, se obtienen fragmentos adaptados. En un primer paso, los fragmentos adaptados se aparean a esferas, donde cada esfera se aparea con un único fragmento adaptado. Al grupo de fragmentos adaptados, se añaden esferas en exceso para asegurar el apareamiento de un único fragmento adaptado por esfera para la mayoría de las esferas (distribución de Poisson).
En un paso siguiente, las esferas se emulsionan en microrreactores de agua en aceite, donde cada microrreactor de agua en aceite comprende una única esfera. Los reactivos de la PCR están presentes en los microrreactores de agua en aceite, lo que permite que se produzca una reacción por PCR en los microrreactores. Posteriormente, los microrreactores se rompen, y las esferas que comprenden ADN (esferas positivas para ADN) se enriquecen. En un paso siguiente, las esferas se cargan en pocillos, donde cada pocillo comprende una única esfera. Los pocillos son preferiblemente parte de una placa PicoTiter™, lo que permite una secuenciación simultánea de una gran cantidad de fragmentos.
Después de la adición de las esferas que llevan enzimas, la secuencia de los fragmentos se determina usando una pirosecuenciación. En pasos sucesivos, la placa PicoTiter™ y las esferas al igual que las esferas enzimáticas incluidas se someten a diferentes desoxirribonucleótidos en presencia de reactivos de secuenciación convencionales y, tras la incorporación de un desoxirribonucleótido, se genera una señal lumínica que se registra. La incorporación del nucleótido correcto generará una señal de pirosecuenciación que puede ser detectada. La pirosecuenciación misma se conoce en la técnica y se describe entre otros en www.biotagebio.com; www.pyrosequencing.com / sección de tecnología. La tecnología se aplica posteriormente, por ejemplo, en la WO 03/004690, la WO 03/054142, la WO 2004/069849, la WO 2004/070005, la WO 2004/070007 y la WO 2005/003375 (todas en nombre de 454 Life Sciences).
En la presente invención, las esferas están equipadas preferiblemente con secuencias (de unión) de cebadores o partes de las mismas que son capaces de unirse a los amplicones, según el caso. En otras formas de realización, los cebadores usados en la amplificación están equipados con secuencias, por ejemplo en su extremo 5', que permiten la unión de los amplicones a las esferas para permitir la polimerización en emulsión posterior seguida de la secuenciación. Alternativamente los amplicones se pueden ligar con adaptadores de secuenciación antes del ligamiento a las esferas o la superficie. Los amplicones secuenciados revelarán la identidad del identificador y así de la presencia o la ausencia del fragmento de restricción en la muestra.
Tecnologías de Solexa
[0054] Uno de los métodos para la secuenciación de alto rendimiento está disponible de Solexa, Reino Unido (www.solexa.co.uk) y se describe, entre otras, en la WO0006770, la WO0027521, la WO058507, la WO0123610, la WO0157248, la WO0157249, la WO02061127, la WO03016565, la WO03048387, la WO2004018497, la WO2004018493, la WO2004050915, la WO2004076692, la WO2005021786, la WO2005047301, la WO2005065814, la WO2005068656, la WO2005068089 y la WO2005078130. En esencia, el método empieza con fragmentos ligados a adaptadores de ADN genómico. El ADN ligado al adaptador se une de forma aleatoria a un manto denso de cebadores que se fijan a una superficie sólida, típicamente en una célula de flujo. El otro extremo del fragmento ligado a un adaptador hibrida con un cebador complementario en la superficie. Los cebadores se extienden en presencia de nucleótidos y polimerasas en una amplificación denominada de puente en fase sólida para proporcionar fragmentos bicatenarios. Esta amplificación de puente en fase sólida puede ser una amplificación selectiva. La desnaturalización y la repetición de la amplificación de puente en fase sólida da lugar a agrupaciones densas de fragmentos amplificados distribuidos sobre la superficie. La secuenciación se inicia por adición de cuatro nucleótidos de terminación reversibles marcados de manera diferente, cebadores y polimerasa a la célula de flujo. Después de la primera ronda de extensión de cebadores, se detectan los marcadores, se registra la identidad de las primeras bases incorporadas y se eliminan el extremo 3' bloqueado y el fluoróforo de la base incorporada. Luego la identidad de la segunda base se determina de la misma manera y así continúa la secuenciación.
[0055] En la presente descripción, los fragmentos de restricción ligados a adaptadores o los amplicones se unen a la superficie a través de la secuencia de unión de cebadores o la secuencia de cebador. La secuencia se determina como se ha resumido, incluida la secuencia identificadora y (parte de) el fragmento de restricción. La tecnología de Solexa disponible actualmente permite la secuenciación de fragmentos de aproximadamente 25 pares de bases. Mediante el diseño económico de los adaptadores y los cebadores unidos a la superficie, el paso de secuenciación lee a través del identificador de muestra, los restos de la secuencia de reconocimiento de la endonucleasa de restricción y cualquier base selectiva opcional. Cuando se usa un identificador de muestra de 6 pb, los restos son del cortador raro EcoRI (AACCT), el uso de dos bases selectivas produce una secuencia interna del fragmento de restricción de 12 pb que se puede usar para identificar únicamente el fragmento de restricción en la muestra.
[0056] En una forma de realización preferida basada en la tecnología de secuenciación de Solexa anterior, la amplificación de los fragmentos de restricción ligados a adaptadores se realiza con un cebador que contiene como mucho un nucleótido selectivo en su extremo 3', preferiblemente ningún nucleótido selectivo en su extremo 3', es decir, el cebador solo es complementario al adaptador (un cebador 0).
[0057] En formas de realización alternativas dirigidas a los métodos de secuenciación descritos aquí, los cebadores usados en la amplificación pueden contener secciones específicas (como alternativa a las secuencias de cebador o de unión de cebadores aquí descritas) que se usan en el paso de secuenciación posterior para unir los fragmentos de restricción cubiertos por un adaptador o amplicones a la superficie. Estos se representan generalmente como la región clave o la secuencia compatible con el cebador 5'.
[0058] En una forma de realización de la presente descripción, la muestra de ácido nucleico se digiere con al menos una enzima de restricción y se liga al menos un adaptador que comprende una secuencia de reconocimiento para una endonucleasa de restricción de tipo IIs. La digestión posterior del fragmento de restricción ligado a un adaptador con una endonucleasa de restricción de tipo IIs produce, ya que la distancia entre el sitio de reconocimiento y el de restricción de una enzima de tipo IIs es relativamente corta (hasta aproximadamente 30 nucleótidos), un fragmento de restricción más corto y uno más largo, a los que puede ligarse un adaptador compatible con el sitio de restricción IIs. Típicamente, se desconoce la proyección del sitio restringido por IIs, de modo que se puede usar un conjunto de adaptadores que están degenerados en la proyección. Después de la amplificación (selectiva), los amplicones se pueden secuenciar. La secuencia de adaptador en esta forma de realización sigue generalmente: 5'-sitio de unión del cebador—secuencia identificadora de muestra—secuencia final cohesiva de tipo IIs degenerada-3'. El cebador de PCR asociado sigue generalmente: secuencia de cebador---secuencia identificadora de muestra—secuencia final cohesiva de tipo IIs degenerada—nucleótidos selectivos-3'.
El cebador usado para iniciar la secuenciación por síntesis tiene entonces generalmente la estructura: 5'-sitio de unión del cebador-3'. Se puede preferir un paso de selección de tamaño después de digerir con la enzima IIs para eliminar los fragmentos más pequeños. Como en esta forma de realización los restos del sitio de restricción para este tipo de enzima tienen típicamente del orden de 2-4 pb, esto da lugar a la combinación con un identificador de muestra de 6 pb en la secuenciación de 15-17 pb de un fragmento de restricción.
[0059] En otro aspecto, la presente descripción se refiere a kits que comprenden uno o más cebadores, y/o uno o más adaptadores para usar en el método, aparte de componentes convencionales para los kits per se. Además, la presente invención encuentra aplicación en, entre otros, el uso del método para la identificación de marcadores moleculares, para la genotipificación, el análisis de segregantes en masa, el mapeo genético, el retrocruce asistido por marcadores, el mapeo de locus de rasgos cuantitativos y el mapeo de desequilibrio de enlace.
Ejemplo
[0060] Se aisló ADN de 2 progenitores y 88 descendientes usando métodos convencionales. Los progenitores (2x) y los descendientes (= 4x) estaban en dúplex con diferentes índices para probar la reproducibilidad. Las etiquetas usadas para distinguir las muestras entre sí diferían al menos en 2 nucleótidos de cualquier otra etiqueta usada en los experimentos. La calidad se está evaluando a lo largo de todos los varios pasos usando geles de agarosa y PAA.
Ejemplo 1
[0061] Para cada muestra de ADN se realiza un paso de restricción-ligamiento usando EcoRI y MseI como enzimas. Los adaptadores se basan en las secuencias de hibridación situadas en la superficie del sistema de secuenciación de alto rendimiento de Solexa, más en particular el adaptador de EcoRI contiene la secuencia P5 (parte del cebador de la secuencia) y el adaptador de MseI contiene la secuencia P7 (secuencia del cebador de la PCR de puente). El adaptador de EcoRI contiene además la etiqueta de identificación de muestra. Se usan 96 adaptadores diferentes de EcoRI y un adaptador de MseI. Es posible usar un adaptador de EcoRI degenerado. La preparación del modelo incluye un paso de selección de tamaño por incubación de la mezcla durante 10 minutos a 80 grados Celsius después del paso de restricción (EcoRI+ MseI) pero antes del paso de ligamiento de adaptadores. Los fragmentos más pequeños de 130 nt se eliminan (en una muestra de maíz).
La complejidad de la mezcla se reduce por una preamplificación selectiva usando cebadores 1 (es decir, con un nucleótido selectivo de forma aleatoria en el extremo 3', usando 96 cebadores EcoRI+1 y un cebador MseI+1 (o un cebador EcoRI+1 con etiqueta degenerada y un cebador MseI+1).
La amplificación selectiva para reducir la complejidad de la mezcla al tamaño deseado se realiza usando cebadores EcoRI+2 (= lado P5) y MseI+3 (= lado P7) que necesitan el uso de 96 cebadores EcoRI+2 y un cebador MseI+3.
La PCR de cola se realiza usando un cebador de EcoRI con la secuencia del cebador de PCR de puente P5 como la cola. Los productos se purifican usando columnas Sephadex™. Se determinan y se normalizan las concentraciones y se crean grupos. Los grupos se someten a la secuenciación masiva en paralelo basándose en la tecnología de Solexa que comprende la amplificación por PCR de puente y la secuenciación seguida de un análisis de datos para determinar los genotipos de los progenitores y los descendientes.
[0062] Un escenario alternativo no usa la PCR de cola, sino que emplea cebadores EcoRI+2 fosforilados. Debido a la falta de coincidencia con el adaptador original, la temperatura de apareamiento en el perfil de amplificación se baja 3 grados Celsius a 13 ciclos de touch-down de 62-53 grados Celsius seguidos de 23 ciclos a 53 grados
Celsius. Después del ligamiento del adaptador con la secuencia de la PCR de puente P5, se realiza la PCR con los cebadores de la PCR de puente P5 y 'P7.
[0063] Un segundo escenario alternativo se basa en la preparación del modelo estándar como se ha resumido aquí antes, la (pre)amplificación selectiva para reducir la complejidad. La amplificación selectiva se realiza con cebadores que contienen los sitios de restricción de EcoRI y MseI reconstituidos. Esto permite la eliminación de las secuencias adaptadoras antes de la secuenciación, reduciendo así la cantidad de datos que hay que analizar. Purificación de los productos por columnas Sephadex™ para eliminar los restos de la Taq ADN polimerasa. Preparación del modelo donde se sustituyen secuencias adaptadoras (de sitio reconstituido) por adaptadores de Solexa usando un adaptador de EcoRI y la enzima EcoRI aumentados diez veces para compensar el mayor número de sitios de EcoRI en comparación con el ADN genómico. Los adaptadores de EcoRI de Solexa también contienen las etiquetas, por lo tanto, se necesitan 96 adaptadores de EcoRI de Solexa etiquetados. La cadena inferior del adaptador está bloqueada en el extremo 3' (en este caso por un amino 3') para bloquear la extensión por una polimerasa. La PCR se realiza con los cebadores de PCR de puente P5 y P7. Los productos se purifican por columnas Qiagen.
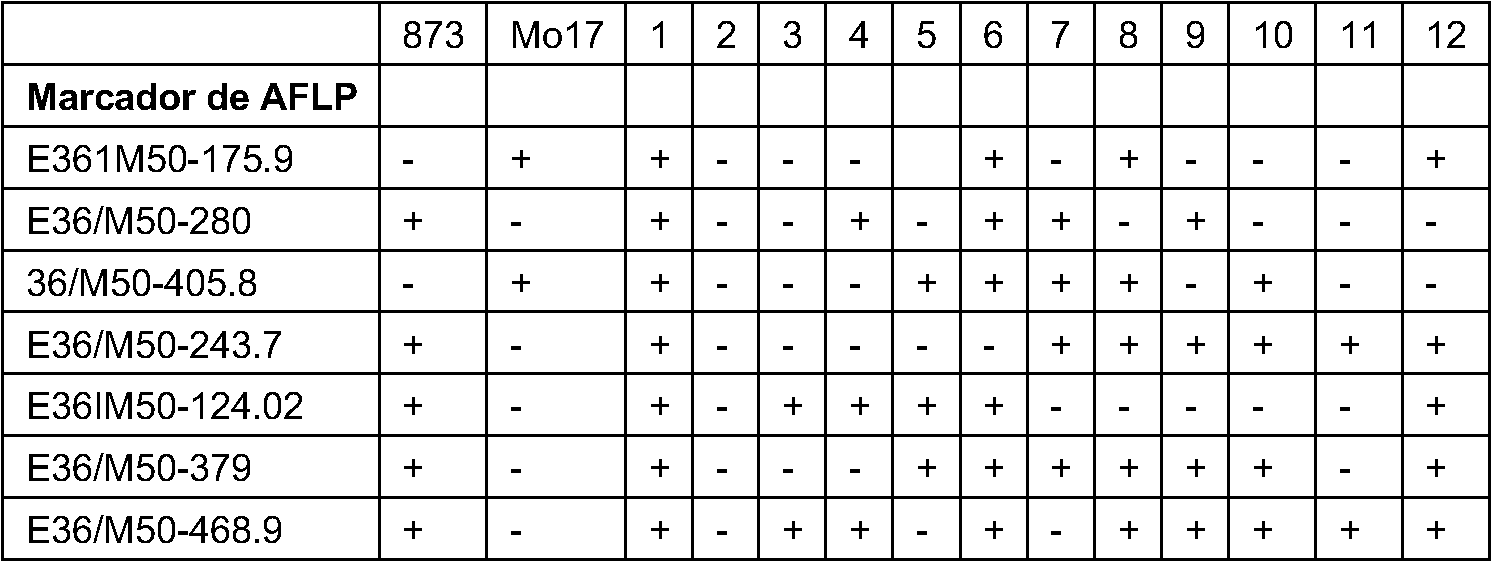
Ejemplo 2:
[0064] La detección basada en secuencias de fragmentos AFLP se realizó usando la tecnología de matriz de moléculas únicas clonal de Solexa (CSMATM), una plataforma de secuenciación por síntesis capaz de analizar hasta 40 millones de fragmentos individuales en una única ronda de secuencia. La secuencia experimental implica la preparación del modelo AFLP, la amplificación selectiva (AFLP), la amplificación de puente de moléculas únicas y la secuenciación de millones de etiquetas de secuencias de un extremo de una enzima de restricción de los fragmentos AFLP. Las líneas parentales de maíz B73 y Mol7 y 87 líneas endogámicas recombinantes (RILs) se usaron y secuenciaron sobre 8,9 millones de terminales de fragmentos AFLP de EcoRI se secuenciaron para proporcionar una prueba de principio para la detección de AFLP basada en secuencias. Se seleccionaron las líneas parentales B73 y Mo17 y 87 RILs. Se prepararon modelos de AFLP usando la combinación de enzimas de restricción EcoRI/MseI. La amplificación selectiva se realizó usando cebadores de AFLP 2/+3. Se prepararon fragmentos modelo para la amplificación de puente CSMA de Solexa realizando una segunda restricción/ligamiento usando adaptadores de EcoRI que contienen secuencias de etiqueta de identificación (ID) de muestra de 5 pb únicas. Las líneas parentales y tres muestras RIL se incluyeron dos veces usando diferentes etiquetas de ID de muestra de 5 pb para medir la reproducibilidad intraexperimental.
Los marcadores de AFLP basados en secuencias fueron identificados por extracción de etiquetas de secuencias de 27 pb observadas con diferentes frecuencias en B73 y Mol7, segregando en los descendientes RIL.
Los datos de marcadores de AFLP basados en secuencias se compararon con puntuaciones de marcadores de AFLP obtenidas por obtención de huellas de AFLP convencional usando la detección basada en la longitud de las cuatro combinaciones de cebadores EcoRI/MseI 3/+3 correspondientes.
Estadísticas de ronda de secuencia 5 células de flujo
[0065]
# etiquetas de secuencia generadas 8.941.407
# etiquetas de secuencia con IDs de muestra conocidas 8.029.595
# etiquetas de secuencia diferentes con IDs de muestra conocidas 206.758 # Mpb de datos de secuencia generados 241,4
rango de frecuencia de # total etiquetas de secuencia por muestra 55.374-112.527
# marcadores de AFLP de etiquetas de secuencia 125
rango de frecuencia de marcadores de AFLP de etiquetas de secuencia en la clasificación de 90-17.218 progenitores presente
Definición y clasificación de marcadores de AFLP de etiquetas de secuencia
[0066]
- tabular la representación por muestra de etiquetas de secuencia
- eliminar las etiquetas de secuencia con IDs de muestra desconocidas
- normalizar la representación de las muestras en función de las etiquetas de secuencia totales por muestra - eliminar las etiquetas de secuencia con una diferencia de frecuencia > 2 veces en los duplicados parentales - promediar las frecuencias de etiqueta de los duplicados parentales
- definir marcador de AFLP de etiqueta de secuencia si la frecuencia P1/P2 excede el valor umbral - clasificar la presencia/ausencia de los marcadores de etiqueta de secuencia en los descendientes RIL Distribución de marcadores de AFLP AFLP 3/+3: secuencia/basados en gel
[0067]
Reproducibilidad de duplicados de marcadores de AFLP de etiqueta secuencia de 3 descendientes RIL [0068]
# marcadores de AFLP de etiquetas de secuencia clasificados 125 # número de puntos de datos en comparación 375 # puntos de datos idénticos para los duplicados 372 % concordancia de duplicados intraexperimentales 99,2%
Detección en gel plano convencional:
Detección basada en Solexa
Claims (21)
1. Método para detectar polimorfismos en fragmentos de restricción de dos o más muestras, que comprende los pasos de:
(a) proporcionar dos o más muestras de ácido nucleico;
(b) digerir cada muestra de ácido nucleico con al menos una endonucleasa de restricción para obtener un conjunto de fragmentos de restricción;
(c) proporcionar adaptadores sintéticos bicatenarios que comprenden
- una secuencia compatible con el cebador 5', seguida de una sección identificadora específica de muestra, y
- un extremo 3' que se puede ligar al extremo romo o saliente de un fragmento de restricción;
(d) ligar los adaptadores sintéticos bicatenarios a los fragmentos de restricción en el conjunto, para proporcionar un conjunto de fragmentos de restricción ligados a adaptadores;
(e) opcionalmente amplificar el conjunto de fragmentos de restricción ligados a adaptadores, con uno o más cebadores que son al menos complementarios a:
- la sección identificadora específica de muestra,
- una sección que es complementaria a los restos de la secuencia de reconocimiento de la endonucleasa de restricción,
para proporcionar fragmentos de restricción ligados a adaptadores amplificados (amplicones);
(f) determinar la secuencia de al menos
- la sección identificadora específica de muestra;
- los restos de la secuencia de reconocimiento de la endonucleasa de restricción; y,
- parte de la secuencia del fragmento de restricción situada adyacente a los restos de la secuencia de reconocimiento de la endonucleasa de restricción; y
(g) detectar polimorfismos en la secuencia de los fragmentos de restricción ligados a adaptadores en las dos o más muestras obtenidas en el paso (f).
2. Método según la reivindicación 1, donde los fragmentos de restricción son marcadores moleculares.
3. Método según la reivindicación 2, donde los marcadores moleculares son marcadores de AFLP.
4. Método según la reivindicación 1, donde dos o más muestras se comparan para la presencia o la ausencia de fragmentos de restricción y/o marcadores moleculares.
5. Método según la reivindicación 1, donde dos o más muestras se combinan en un grupo después del paso de ligar los adaptadores.
6. Método según la reivindicación 5, donde para cada muestra en el grupo se usa un identificador específico de muestra que difiere de los otros identificadores específicos de muestra en el grupo.
7. Método según la reivindicación 1, donde los cebadores contienen uno o más nucleótidos selectivos en el extremo 3'.
8. Método según la reivindicación 1, donde la endonucleasa de restricción es una endonucleasa de restricción de tipo II.
9. Método según la reivindicación 1, donde la endonucleasa de restricción es una endonucleasa de restricción de tipo IIs.
10. Método según la reivindicación 1, donde se usan dos o más endonucleasas de restricción.
11. Método según la reivindicación 1, donde se realiza la secuenciación por medio de secuenciación de alto rendimiento.
12. Método según la reivindicación 11, donde la secuenciación de alto rendimiento se realiza en un soporte sólido.
13. Método según la reivindicación 11, donde la secuenciación de alto rendimiento se basa en la secuenciación por síntesis.
14. Método según la reivindicación 11, donde la secuenciación de alto rendimiento comprende los pasos de:
- aparear los amplicones o fragmentos de restricción ligados a adaptadores a esferas, donde cada esfera se aparea con un único fragmento de restricción ligado a un adaptador o amplicón;
- emulsionar las esferas en microrreactores de agua en aceite, donde cada microrreactor de agua en aceite comprende una única esfera;
- realizar una PCR en emulsión para amplificar fragmentos de restricción ligados a adaptadores o amplicones en la superficie de las esferas;
- opcionalmente, seleccionar/enriquecer las esferas que contienen amplicones amplificados;
- cargar las esferas en pocillos, donde cada pocillo comprende una única esfera; y
- determinar la secuencia de nucleótidos de los fragmentos de restricción ligados a adaptadores amplificados o amplicones amplificados usando la generación de una señal de pirofosfato.
15. Método según la reivindicación 11, donde la secuenciación de alto rendimiento comprende los pasos de:
- aparear los fragmentos de restricción ligados a adaptadores o amplicones a una superficie que contiene primeros y segundos cebadores o primeras y segundas secuencias de unión de cebadores respectivamente, - realizar una amplificación de puente para proporcionar agrupaciones de fragmentos de restricción ligados a adaptadores amplificados o amplicones amplificados,
- determinar la secuencia de nucleótidos de los fragmentos de restricción ligados a adaptadores amplificados o amplicones amplificados usando nucleótidos de terminación reversibles marcados.
16. Método según la reivindicación 1, donde el identificador tiene de 4 a 16 pb, preferiblemente de 4 a 10, más preferiblemente de 4 a 8, más preferiblemente de 4 a 6 pb.
17. Método según la reivindicación 1, donde el identificador no contiene 2 o más bases consecutivas idénticas.
18. Método según la reivindicación 1, donde, para dos o más muestras, los identificadores correspondientes contienen al menos dos nucleótidos diferentes.
19. Uso del método de cualquiera de las reivindicaciones 1-18 para la identificación de marcadores moleculares, para la genotipificación, el análisis de segregantes en masa, el mapeo genético, el retrocruce asistido por marcadores, el mapeo de locus de rasgos cuantitativos, el mapeo de desequilibrio de enlace.
20. Kit para usar en el método según cualquiera de las reivindicaciones 1-18 que comprende:
- uno o más adaptadores que comprenden una secuencia compatible con el cebador 5', seguida de una sección identificadora específica de muestra, y un extremo 3' de que se puede ligar al extremo romo o saliente de un fragmento de restricción para proporcionar fragmentos de restricción ligados a adaptadores, y - uno o más cebadores capaces de hibridar con los fragmentos de restricción ligados a adaptadores, que comprenden una parte que es complementaria al uno o más adaptadores y que comprenden en el extremo 3' uno o más nucleótidos selectivos para proporcionar un subconjunto de fragmentos de restricción ligados a adaptadores amplificados por amplificación selectiva.
21. Kit para usar en el método según cualquiera de las reivindicaciones 1-18 que comprende:
- uno o más adaptadores que comprenden una secuencia compatible con el cebador 5', seguida de una sección identificadora específica de muestra, y un extremo 3' que se puede ligar al extremo romo o saliente de un fragmento de restricción para proporcionar fragmentos de restricción ligados a adaptadores, y - uno o más cebadores capaces de hibridar con los fragmentos de restricción ligados a adaptadores, que comprenden una parte que es complementaria al uno o más adaptadores y que comprenden en el extremo 3' uno o más nucleótidos selectivos para proporcionar un subconjunto de fragmentos de restricción ligados a adaptadores amplificados por amplificación selectiva, y
- uno o más cebadores para la amplificación no selectiva que son complementarios al uno o más adaptadores solo.
Applications Claiming Priority (2)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US78870606P | 2006-04-04 | 2006-04-04 | |
| US88005207P | 2007-01-12 | 2007-01-12 |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| ES2829295T3 true ES2829295T3 (es) | 2021-05-31 |
Family
ID=38508899
Family Applications (3)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| ES17163116T Active ES2829295T3 (es) | 2006-04-04 | 2007-04-04 | Detección de alto rendimiento de marcadores moleculares basados en AFLP y secuenciación de alto rendimiento |
| ES07747276.9T Active ES2545264T3 (es) | 2006-04-04 | 2007-04-04 | Detección de alto rendimiento de marcadores moleculares basada en fragmentos de restricción |
| ES15158621.1T Active ES2645661T3 (es) | 2006-04-04 | 2007-04-04 | Detección de alto rendimiento de marcadores moleculares basada en fragmentos de restricción |
Family Applications After (2)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| ES07747276.9T Active ES2545264T3 (es) | 2006-04-04 | 2007-04-04 | Detección de alto rendimiento de marcadores moleculares basada en fragmentos de restricción |
| ES15158621.1T Active ES2645661T3 (es) | 2006-04-04 | 2007-04-04 | Detección de alto rendimiento de marcadores moleculares basada en fragmentos de restricción |
Country Status (8)
| Country | Link |
|---|---|
| US (6) | US20090253581A1 (es) |
| EP (3) | EP2963127B1 (es) |
| JP (2) | JP5389638B2 (es) |
| DK (2) | DK2002017T3 (es) |
| ES (3) | ES2829295T3 (es) |
| PL (1) | PL2963127T3 (es) |
| PT (1) | PT2963127T (es) |
| WO (1) | WO2007114693A2 (es) |
Families Citing this family (133)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2006122215A2 (en) | 2005-05-10 | 2006-11-16 | State Of Oregon Acting By & Through The State Board Of Higher Education On Behalf Of The University Of Oregon | Methods of mapping polymorphisms and polymorphism microarrays |
| EP2302070B1 (en) | 2005-06-23 | 2012-08-22 | Keygene N.V. | Strategies for high throughput identification and detection of polymorphisms |
| CA2623539C (en) | 2005-09-29 | 2015-12-15 | Keygene N.V. | High throughput screening of mutagenized populations |
| US10316364B2 (en) | 2005-09-29 | 2019-06-11 | Keygene N.V. | Method for identifying the source of an amplicon |
| DK2789696T3 (en) | 2005-12-22 | 2016-02-29 | Keygene Nv | A method for high-throughput AFLP-based polymorphism |
| US9365901B2 (en) | 2008-11-07 | 2016-06-14 | Adaptive Biotechnologies Corp. | Monitoring immunoglobulin heavy chain evolution in B-cell acute lymphoblastic leukemia |
| US8748103B2 (en) | 2008-11-07 | 2014-06-10 | Sequenta, Inc. | Monitoring health and disease status using clonotype profiles |
| US8691510B2 (en) | 2008-11-07 | 2014-04-08 | Sequenta, Inc. | Sequence analysis of complex amplicons |
| US9506119B2 (en) | 2008-11-07 | 2016-11-29 | Adaptive Biotechnologies Corp. | Method of sequence determination using sequence tags |
| US8628927B2 (en) | 2008-11-07 | 2014-01-14 | Sequenta, Inc. | Monitoring health and disease status using clonotype profiles |
| US9528160B2 (en) | 2008-11-07 | 2016-12-27 | Adaptive Biotechnolgies Corp. | Rare clonotypes and uses thereof |
| US8236503B2 (en) | 2008-11-07 | 2012-08-07 | Sequenta, Inc. | Methods of monitoring conditions by sequence analysis |
| US8685898B2 (en) | 2009-01-15 | 2014-04-01 | Imdaptive, Inc. | Adaptive immunity profiling and methods for generation of monoclonal antibodies |
| CN102369298B (zh) * | 2009-01-30 | 2017-03-22 | 牛津纳米孔技术有限公司 | 跨膜测序中用于核酸构建体的衔接体 |
| GB2472371B (en) * | 2009-04-24 | 2011-10-26 | Selectamark Security Systems Plc | Synthetic nucleotide containing compositions for use in security marking of property and/or for marking a thief or attacker |
| US9085798B2 (en) | 2009-04-30 | 2015-07-21 | Prognosys Biosciences, Inc. | Nucleic acid constructs and methods of use |
| EP2248914A1 (en) * | 2009-05-05 | 2010-11-10 | Max-Planck-Gesellschaft zur Förderung der Wissenschaften e.V. | The use of class IIB restriction endonucleases in 2nd generation sequencing applications |
| CA2765949C (en) | 2009-06-25 | 2016-03-29 | Fred Hutchinson Cancer Research Center | Method of measuring adaptive immunity |
| US9043160B1 (en) | 2009-11-09 | 2015-05-26 | Sequenta, Inc. | Method of determining clonotypes and clonotype profiles |
| US20190300945A1 (en) | 2010-04-05 | 2019-10-03 | Prognosys Biosciences, Inc. | Spatially Encoded Biological Assays |
| HRP20151294T1 (hr) | 2010-04-05 | 2016-03-11 | Prognosys Biosciences, Inc. | Biološka testiranja sa prostornim kodiranjem |
| US10787701B2 (en) | 2010-04-05 | 2020-09-29 | Prognosys Biosciences, Inc. | Spatially encoded biological assays |
| US8766054B2 (en) | 2010-12-08 | 2014-07-01 | Hm.Clause | Phytophthora resistance in sweet peppers |
| GB201106254D0 (en) | 2011-04-13 | 2011-05-25 | Frisen Jonas | Method and product |
| CN103827320B (zh) | 2011-07-25 | 2017-04-26 | 牛津纳米孔技术有限公司 | 使用跨膜孔进行双链多核苷酸测序的发夹环方法 |
| US10385475B2 (en) | 2011-09-12 | 2019-08-20 | Adaptive Biotechnologies Corp. | Random array sequencing of low-complexity libraries |
| WO2013059725A1 (en) | 2011-10-21 | 2013-04-25 | Adaptive Biotechnologies Corporation | Quantification of adaptive immune cell genomes in a complex mixture of cells |
| EP3388535B1 (en) | 2011-12-09 | 2021-03-24 | Adaptive Biotechnologies Corporation | Diagnosis of lymphoid malignancies and minimal residual disease detection |
| US9499865B2 (en) | 2011-12-13 | 2016-11-22 | Adaptive Biotechnologies Corp. | Detection and measurement of tissue-infiltrating lymphocytes |
| CN104334739A (zh) * | 2012-01-13 | 2015-02-04 | Data生物有限公司 | 通过新一代测序进行基因分型 |
| EP2814959B1 (en) | 2012-02-17 | 2018-01-17 | Fred Hutchinson Cancer Research Center | Compositions and methods for accurately identifying mutations |
| US10077478B2 (en) | 2012-03-05 | 2018-09-18 | Adaptive Biotechnologies Corp. | Determining paired immune receptor chains from frequency matched subunits |
| HUE051845T2 (hu) | 2012-03-20 | 2021-03-29 | Univ Washington Through Its Center For Commercialization | Módszerek a tömegesen párhuzamos DNS-szekvenálás hibaarányának csökkentésére duplex konszenzus szekvenálással |
| MX354329B (es) | 2012-05-08 | 2018-02-27 | Adaptive Biotechnologies Corp | Composiciones y metodos para medir y calibrar el error de amplificacion en reacciones de pcr multiple. |
| ES2749118T3 (es) | 2012-10-01 | 2020-03-19 | Adaptive Biotechnologies Corp | Evaluación de la inmunocompetencia por la diversidad de los receptores de inmunidad adaptativa y caracterización de la clonalidad |
| EP3511423B2 (en) | 2012-10-17 | 2024-05-29 | Spatial Transcriptomics AB | Methods and product for optimising localised or spatial detection of gene expression in a tissue sample |
| WO2015160439A2 (en) | 2014-04-17 | 2015-10-22 | Adaptive Biotechnologies Corporation | Quantification of adaptive immune cell genomes in a complex mixture of cells |
| GB201314695D0 (en) | 2013-08-16 | 2013-10-02 | Oxford Nanopore Tech Ltd | Method |
| CN105209634B (zh) | 2013-03-08 | 2020-05-12 | 牛津纳米孔技术公司 | 酶停滞方法 |
| LT3013984T (lt) | 2013-06-25 | 2023-06-12 | Prognosys Biosciences, Inc. | Biologinių taikinių erdvinių modelių mėginyje nustatymo būdai |
| US9708657B2 (en) | 2013-07-01 | 2017-07-18 | Adaptive Biotechnologies Corp. | Method for generating clonotype profiles using sequence tags |
| WO2015134787A2 (en) | 2014-03-05 | 2015-09-11 | Adaptive Biotechnologies Corporation | Methods using randomer-containing synthetic molecules |
| US11390921B2 (en) | 2014-04-01 | 2022-07-19 | Adaptive Biotechnologies Corporation | Determining WT-1 specific T cells and WT-1 specific T cell receptors (TCRs) |
| US10066265B2 (en) | 2014-04-01 | 2018-09-04 | Adaptive Biotechnologies Corp. | Determining antigen-specific t-cells |
| GB201418159D0 (en) | 2014-10-14 | 2014-11-26 | Oxford Nanopore Tech Ltd | Method |
| ES2784343T3 (es) | 2014-10-29 | 2020-09-24 | Adaptive Biotechnologies Corp | Detección simultánea altamente multiplexada de ácidos nucleicos que codifican heterodímeros de receptores inmunes adaptativos emparejados de muchas muestras |
| US10246701B2 (en) | 2014-11-14 | 2019-04-02 | Adaptive Biotechnologies Corp. | Multiplexed digital quantitation of rearranged lymphoid receptors in a complex mixture |
| AU2015353581A1 (en) | 2014-11-25 | 2017-06-15 | Adaptive Biotechnologies Corporation | Characterization of adaptive immune response to vaccination or infection using immune repertoire sequencing |
| US11047008B2 (en) | 2015-02-24 | 2021-06-29 | Adaptive Biotechnologies Corporation | Methods for diagnosing infectious disease and determining HLA status using immune repertoire sequencing |
| EP3277294B1 (en) | 2015-04-01 | 2024-05-15 | Adaptive Biotechnologies Corp. | Method of identifying human compatible t cell receptors specific for an antigenic target |
| CN120425026A (zh) | 2015-04-10 | 2025-08-05 | 十程基因技术瑞典公司 | 生物样本的空间区别、多重核酸分析 |
| CA3006792A1 (en) | 2015-12-08 | 2017-06-15 | Twinstrand Biosciences, Inc. | Improved adapters, methods, and compositions for duplex sequencing |
| CN105441572B (zh) * | 2016-01-07 | 2019-10-22 | 南京中医药大学 | 鉴别当归早薹的dna分子标记及其应用 |
| GB201609220D0 (en) | 2016-05-25 | 2016-07-06 | Oxford Nanopore Tech Ltd | Method |
| JP6515884B2 (ja) | 2016-06-29 | 2019-05-22 | トヨタ自動車株式会社 | Dnaプローブの作製方法及びdnaプローブを用いたゲノムdna解析方法 |
| JP7343264B2 (ja) | 2016-06-29 | 2023-09-12 | トヨタ自動車株式会社 | Dnaライブラリーの作製方法及びdnaライブラリーを用いたゲノムdna解析方法 |
| US10428325B1 (en) | 2016-09-21 | 2019-10-01 | Adaptive Biotechnologies Corporation | Identification of antigen-specific B cell receptors |
| EP4053294A1 (en) * | 2017-03-24 | 2022-09-07 | Life Technologies Corporation | Polynucleotide adapters and methods of use thereof |
| WO2018183942A1 (en) | 2017-03-31 | 2018-10-04 | Grail, Inc. | Improved library preparation and use thereof for sequencing-based error correction and/or variant identification |
| JP7056012B2 (ja) | 2017-05-19 | 2022-04-19 | トヨタ自動車株式会社 | ランダムプライマーセット、及びこれを用いたdnaライブラリーの作製方法 |
| SG11202003885UA (en) | 2017-11-08 | 2020-05-28 | Twinstrand Biosciences Inc | Reagents and adapters for nucleic acid sequencing and methods for making such reagents and adapters |
| US11254980B1 (en) | 2017-11-29 | 2022-02-22 | Adaptive Biotechnologies Corporation | Methods of profiling targeted polynucleotides while mitigating sequencing depth requirements |
| WO2019121603A1 (en) | 2017-12-18 | 2019-06-27 | Keygene N.V. | Chemical mutagenesis of cassava |
| JP7047373B2 (ja) | 2017-12-25 | 2022-04-05 | トヨタ自動車株式会社 | 次世代シーケンサー用プライマー並びにその製造方法、次世代シーケンサー用プライマーを用いたdnaライブラリー並びにその製造方法、及びdnaライブラリーを用いたゲノムdna解析方法 |
| GB201807793D0 (en) | 2018-05-14 | 2018-06-27 | Oxford Nanopore Tech Ltd | Method |
| BR112021000409A2 (pt) | 2018-07-12 | 2021-04-06 | Twinstrand Biosciences, Inc. | Métodos e reagentes para caracterizar edição genômica, expansão clonal e aplicações associadas |
| US11519033B2 (en) | 2018-08-28 | 2022-12-06 | 10X Genomics, Inc. | Method for transposase-mediated spatial tagging and analyzing genomic DNA in a biological sample |
| CN113767176A (zh) | 2018-12-10 | 2021-12-07 | 10X基因组学有限公司 | 成像系统硬件 |
| US11926867B2 (en) | 2019-01-06 | 2024-03-12 | 10X Genomics, Inc. | Generating capture probes for spatial analysis |
| US11649485B2 (en) | 2019-01-06 | 2023-05-16 | 10X Genomics, Inc. | Generating capture probes for spatial analysis |
| WO2020169830A1 (en) | 2019-02-21 | 2020-08-27 | Keygene N.V. | Genotyping of polyploids |
| WO2020243579A1 (en) | 2019-05-30 | 2020-12-03 | 10X Genomics, Inc. | Methods of detecting spatial heterogeneity of a biological sample |
| WO2021091611A1 (en) | 2019-11-08 | 2021-05-14 | 10X Genomics, Inc. | Spatially-tagged analyte capture agents for analyte multiplexing |
| WO2021092433A2 (en) | 2019-11-08 | 2021-05-14 | 10X Genomics, Inc. | Enhancing specificity of analyte binding |
| EP4073245A1 (en) | 2019-12-12 | 2022-10-19 | Keygene N.V. | Semi-solid state nucleic acid manipulation |
| WO2021123062A1 (en) | 2019-12-20 | 2021-06-24 | Keygene N.V. | Ngs library preparation using covalently closed nucleic acid molecule ends |
| SG11202106899SA (en) | 2019-12-23 | 2021-09-29 | 10X Genomics Inc | Methods for spatial analysis using rna-templated ligation |
| CN115038794A (zh) | 2019-12-23 | 2022-09-09 | 10X基因组学有限公司 | 在基于分区的测定中使用固定生物样品的组合物和方法 |
| US12365942B2 (en) | 2020-01-13 | 2025-07-22 | 10X Genomics, Inc. | Methods of decreasing background on a spatial array |
| US12405264B2 (en) | 2020-01-17 | 2025-09-02 | 10X Genomics, Inc. | Electrophoretic system and method for analyte capture |
| US11702693B2 (en) | 2020-01-21 | 2023-07-18 | 10X Genomics, Inc. | Methods for printing cells and generating arrays of barcoded cells |
| US11732299B2 (en) | 2020-01-21 | 2023-08-22 | 10X Genomics, Inc. | Spatial assays with perturbed cells |
| US20210230681A1 (en) | 2020-01-24 | 2021-07-29 | 10X Genomics, Inc. | Methods for spatial analysis using proximity ligation |
| US11821035B1 (en) | 2020-01-29 | 2023-11-21 | 10X Genomics, Inc. | Compositions and methods of making gene expression libraries |
| US12076701B2 (en) | 2020-01-31 | 2024-09-03 | 10X Genomics, Inc. | Capturing oligonucleotides in spatial transcriptomics |
| US11898205B2 (en) | 2020-02-03 | 2024-02-13 | 10X Genomics, Inc. | Increasing capture efficiency of spatial assays |
| US12110541B2 (en) | 2020-02-03 | 2024-10-08 | 10X Genomics, Inc. | Methods for preparing high-resolution spatial arrays |
| US11732300B2 (en) | 2020-02-05 | 2023-08-22 | 10X Genomics, Inc. | Increasing efficiency of spatial analysis in a biological sample |
| US12129516B2 (en) | 2020-02-07 | 2024-10-29 | 10X Genomics, Inc. | Quantitative and automated permeabilization performance evaluation for spatial transcriptomics |
| US11835462B2 (en) | 2020-02-11 | 2023-12-05 | 10X Genomics, Inc. | Methods and compositions for partitioning a biological sample |
| US12399123B1 (en) | 2020-02-14 | 2025-08-26 | 10X Genomics, Inc. | Spatial targeting of analytes |
| US12281357B1 (en) | 2020-02-14 | 2025-04-22 | 10X Genomics, Inc. | In situ spatial barcoding |
| US11211144B2 (en) | 2020-02-18 | 2021-12-28 | Tempus Labs, Inc. | Methods and systems for refining copy number variation in a liquid biopsy assay |
| US11211147B2 (en) | 2020-02-18 | 2021-12-28 | Tempus Labs, Inc. | Estimation of circulating tumor fraction using off-target reads of targeted-panel sequencing |
| US11475981B2 (en) | 2020-02-18 | 2022-10-18 | Tempus Labs, Inc. | Methods and systems for dynamic variant thresholding in a liquid biopsy assay |
| US11891654B2 (en) | 2020-02-24 | 2024-02-06 | 10X Genomics, Inc. | Methods of making gene expression libraries |
| US11926863B1 (en) | 2020-02-27 | 2024-03-12 | 10X Genomics, Inc. | Solid state single cell method for analyzing fixed biological cells |
| US11768175B1 (en) | 2020-03-04 | 2023-09-26 | 10X Genomics, Inc. | Electrophoretic methods for spatial analysis |
| CN111304341A (zh) * | 2020-04-10 | 2020-06-19 | 西北民族大学 | 一种鉴定食品中猪源性成分的pcr-aflp方法 |
| EP4139485B1 (en) | 2020-04-22 | 2023-09-06 | 10X Genomics, Inc. | Methods for spatial analysis using targeted rna depletion |
| WO2021236625A1 (en) | 2020-05-19 | 2021-11-25 | 10X Genomics, Inc. | Electrophoresis cassettes and instrumentation |
| EP4414459B1 (en) | 2020-05-22 | 2025-09-03 | 10X Genomics, Inc. | Simultaneous spatio-temporal measurement of gene expression and cellular activity |
| AU2021275906A1 (en) | 2020-05-22 | 2022-12-22 | 10X Genomics, Inc. | Spatial analysis to detect sequence variants |
| WO2021242834A1 (en) | 2020-05-26 | 2021-12-02 | 10X Genomics, Inc. | Method for resetting an array |
| EP4600376A3 (en) | 2020-06-02 | 2025-10-22 | 10X Genomics, Inc. | Spatial transcriptomics for antigen-receptors |
| EP4025692A2 (en) | 2020-06-02 | 2022-07-13 | 10X Genomics, Inc. | Nucleic acid library methods |
| US12265079B1 (en) | 2020-06-02 | 2025-04-01 | 10X Genomics, Inc. | Systems and methods for detecting analytes from captured single biological particles |
| US12031177B1 (en) | 2020-06-04 | 2024-07-09 | 10X Genomics, Inc. | Methods of enhancing spatial resolution of transcripts |
| EP4421186B1 (en) | 2020-06-08 | 2025-08-13 | 10X Genomics, Inc. | Methods of determining a surgical margin and methods of use thereof |
| US12435363B1 (en) | 2020-06-10 | 2025-10-07 | 10X Genomics, Inc. | Materials and methods for spatial transcriptomics |
| AU2021288090A1 (en) | 2020-06-10 | 2023-01-19 | 10X Genomics, Inc. | Fluid delivery methods |
| EP4446430A3 (en) | 2020-06-10 | 2024-12-18 | 10X Genomics, Inc. | Methods for determining a location of an analyte in a biological sample |
| WO2021263111A1 (en) | 2020-06-25 | 2021-12-30 | 10X Genomics, Inc. | Spatial analysis of dna methylation |
| US11761038B1 (en) | 2020-07-06 | 2023-09-19 | 10X Genomics, Inc. | Methods for identifying a location of an RNA in a biological sample |
| US11981960B1 (en) | 2020-07-06 | 2024-05-14 | 10X Genomics, Inc. | Spatial analysis utilizing degradable hydrogels |
| US12209280B1 (en) | 2020-07-06 | 2025-01-28 | 10X Genomics, Inc. | Methods of identifying abundance and location of an analyte in a biological sample using second strand synthesis |
| US11981958B1 (en) | 2020-08-20 | 2024-05-14 | 10X Genomics, Inc. | Methods for spatial analysis using DNA capture |
| ES2993269T3 (en) | 2020-09-18 | 2024-12-26 | 10X Genomics Inc | Sample handling apparatus and image registration methods |
| US11926822B1 (en) | 2020-09-23 | 2024-03-12 | 10X Genomics, Inc. | Three-dimensional spatial analysis |
| US11827935B1 (en) | 2020-11-19 | 2023-11-28 | 10X Genomics, Inc. | Methods for spatial analysis using rolling circle amplification and detection probes |
| AU2021409136A1 (en) | 2020-12-21 | 2023-06-29 | 10X Genomics, Inc. | Methods, compositions, and systems for capturing probes and/or barcodes |
| EP4421491A3 (en) | 2021-02-19 | 2024-11-27 | 10X Genomics, Inc. | Method of using a modular assay support device |
| EP4301870B1 (en) | 2021-03-18 | 2025-01-15 | 10X Genomics, Inc. | Multiplex capture of gene and protein expression from a biological sample |
| WO2022221425A1 (en) | 2021-04-14 | 2022-10-20 | 10X Genomics, Inc. | Methods of measuring mislocalization of an analyte |
| WO2022236054A1 (en) | 2021-05-06 | 2022-11-10 | 10X Genomics, Inc. | Methods for increasing resolution of spatial analysis |
| ES3030033T3 (en) | 2021-06-03 | 2025-06-26 | 10X Genomics Inc | Methods, compositions, kits, and systems for enhancing analyte capture for spatial analysis |
| EP4196605B1 (en) | 2021-09-01 | 2024-12-04 | 10X Genomics, Inc. | Methods for blocking a capture probe on a spatial array |
| WO2023086880A1 (en) | 2021-11-10 | 2023-05-19 | 10X Genomics, Inc. | Methods, compositions, and kits for determining the location of an analyte in a biological sample |
| EP4305195A2 (en) | 2021-12-01 | 2024-01-17 | 10X Genomics, Inc. | Methods, compositions, and systems for improved in situ detection of analytes and spatial analysis |
| WO2023122033A1 (en) | 2021-12-20 | 2023-06-29 | 10X Genomics, Inc. | Self-test for pathology/histology slide imaging device |
| WO2024121354A1 (en) | 2022-12-08 | 2024-06-13 | Keygene N.V. | Duplex sequencing with covalently closed dna ends |
| WO2024209000A1 (en) | 2023-04-04 | 2024-10-10 | Keygene N.V. | Linkers for duplex sequencing |
| WO2025257212A1 (en) | 2024-06-11 | 2025-12-18 | Keygene N.V. | Screening and regeneration of protoplast callus |
Family Cites Families (85)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO1990008821A1 (en) | 1989-01-31 | 1990-08-09 | University Of Miami | Microdissection and amplification of chromosomal dna |
| IL103267A (en) * | 1991-09-24 | 2004-07-25 | Keygene Nv | Process and kit for amplification of restriction fragment obtained from starting dna |
| US20100267023A1 (en) * | 1992-09-24 | 2010-10-21 | Keygene N.V. | Selective restriction fragment amplification: fingerprinting |
| AU1682595A (en) | 1994-01-21 | 1995-08-08 | North Carolina State University | Methods for within family selection in woody perennials using genetic markers |
| EG23907A (en) | 1994-08-01 | 2007-12-30 | Delta & Pine Land Co | Control of plant gene expression |
| US6013445A (en) | 1996-06-06 | 2000-01-11 | Lynx Therapeutics, Inc. | Massively parallel signature sequencing by ligation of encoded adaptors |
| EP0804618B1 (en) * | 1994-11-28 | 1999-01-27 | E.I. Du Pont De Nemours And Company | Compound microsatellite primers for the detection of genetic polymorphisms |
| US5565340A (en) | 1995-01-27 | 1996-10-15 | Clontech Laboratories, Inc. | Method for suppressing DNA fragment amplification during PCR |
| AU6024598A (en) * | 1997-01-10 | 1998-08-03 | Pioneer Hi-Bred International, Inc. | Hybridization-based genetic amplification and analysis |
| US6090556A (en) | 1997-04-07 | 2000-07-18 | Japan Science & Technology Corporation | Method for quantitatively determining the expression of a gene |
| JP2001515362A (ja) | 1997-05-13 | 2001-09-18 | ディスプレイ・システムズ・バイオテック・アクティーゼルスカブ | mRNAをクローニングする方法および差次的に発現された転写物のディスプレイ(DODET) |
| ES2290979T3 (es) | 1997-12-15 | 2008-02-16 | Csl Behring Gmbh | Cebador marcado para uso en la deteccion de acidos nucleicos diana. |
| CA2273616A1 (en) | 1998-06-08 | 1999-12-08 | The Board Of Trustees Of The Leland Stanford Junior University | Method for parallel screening of allelic variation |
| ATE244771T1 (de) | 1998-07-29 | 2003-07-15 | Keygene Nv | Verfahren zur erkennung von nukleinsäuremethylierungen durch aflp |
| JP2002521064A (ja) | 1998-07-30 | 2002-07-16 | ソレックサ リミテッド | アレイ生体分子およびシークエンシングにおけるその使用 |
| GB0002310D0 (en) | 2000-02-01 | 2000-03-22 | Solexa Ltd | Polynucleotide sequencing |
| US6232067B1 (en) * | 1998-08-17 | 2001-05-15 | The Perkin-Elmer Corporation | Adapter directed expression analysis |
| US6703228B1 (en) | 1998-09-25 | 2004-03-09 | Massachusetts Institute Of Technology | Methods and products related to genotyping and DNA analysis |
| EP1001037A3 (en) | 1998-09-28 | 2003-10-01 | Whitehead Institute For Biomedical Research | Pre-selection and isolation of single nucleotide polymorphisms |
| EP1124990B1 (en) | 1998-10-27 | 2006-01-18 | Affymetrix, Inc. | Complexity management and analysis of genomic dna |
| US6480791B1 (en) * | 1998-10-28 | 2002-11-12 | Michael P. Strathmann | Parallel methods for genomic analysis |
| WO2000027521A1 (en) | 1998-11-06 | 2000-05-18 | Solexa Ltd. | A method for reproducing molecular arrays |
| CA2356697C (en) | 1999-01-06 | 2010-06-22 | Cornell Research Foundation, Inc. | Accelerating identification of single nucleotide polymorphisms and alignment of clones in genomic sequencing |
| US20040029155A1 (en) | 1999-01-08 | 2004-02-12 | Curagen Corporation | Method for identifying a biomolecule |
| DE19911130A1 (de) | 1999-03-12 | 2000-09-21 | Hager Joerg | Verfahren zur Identifikation chromosomaler Regionen und Gene |
| WO2000058507A1 (en) | 1999-03-30 | 2000-10-05 | Solexa Ltd. | Polynucleotide sequencing |
| US6261782B1 (en) * | 1999-04-06 | 2001-07-17 | Yale University | Fixed address analysis of sequence tags |
| DE60038109T2 (de) | 1999-04-09 | 2008-11-27 | Keygene N.V. | Verfahren zur Analyse von AFLP Reaktionsmischungen unter Verwendung von Primer Verlängerungstechniken |
| US20020119448A1 (en) | 1999-06-23 | 2002-08-29 | Joseph A. Sorge | Methods of enriching for and identifying polymorphisms |
| US20030204075A9 (en) | 1999-08-09 | 2003-10-30 | The Snp Consortium | Identification and mapping of single nucleotide polymorphisms in the human genome |
| AU7712400A (en) | 1999-09-23 | 2001-04-24 | Gene Logic, Inc. | Indexing populations |
| EP1218543A2 (en) | 1999-09-29 | 2002-07-03 | Solexa Ltd. | Polynucleotide sequencing |
| US6287778B1 (en) | 1999-10-19 | 2001-09-11 | Affymetrix, Inc. | Allele detection using primer extension with sequence-coded identity tags |
| US6958225B2 (en) | 1999-10-27 | 2005-10-25 | Affymetrix, Inc. | Complexity management of genomic DNA |
| WO2001038572A1 (fr) | 1999-11-19 | 2001-05-31 | Takara Bio Inc. | Methode d'amplification d'acides nucleiques |
| GB0002389D0 (en) | 2000-02-02 | 2000-03-22 | Solexa Ltd | Molecular arrays |
| WO2001075167A1 (en) | 2000-03-31 | 2001-10-11 | Fred Hutchinson Cancer Research Center | Reverse genetic strategy for identifying functional mutations in genes of known sequence |
| EP1282729A2 (en) | 2000-05-15 | 2003-02-12 | Keygene N.V. | Microsatellite-aflp |
| US7300751B2 (en) | 2000-06-30 | 2007-11-27 | Syngenta Participations Ag | Method for identification of genetic markers |
| WO2002057491A2 (en) | 2000-10-24 | 2002-07-25 | The Board Of Trustees Of The Leland Stanford Junior University | Direct multiplex characterization of genomic dna |
| AU2002231934A1 (en) | 2001-01-30 | 2002-08-12 | Solexa Ltd. | Arrayed polynucleotides and their use in genome analysis |
| US20040053236A1 (en) * | 2001-03-30 | 2004-03-18 | Mccallum Claire M. | Reverse genetic strategy for identifying functional mutations in genes of known sequences |
| CA2444994A1 (en) | 2001-04-20 | 2002-10-31 | Karolinska Innovations Ab | Methods for high throughput genome analysis using restriction site tagged microarrays |
| WO2003004690A2 (en) | 2001-07-06 | 2003-01-16 | 454$m(3) CORPORATION | Method for isolation of independent, parallel chemical micro-reactions using a porous filter |
| GB0119719D0 (en) | 2001-08-13 | 2001-10-03 | Solexa Ltd | DNA sequence analysis |
| US6902921B2 (en) | 2001-10-30 | 2005-06-07 | 454 Corporation | Sulfurylase-luciferase fusion proteins and thermostable sulfurylase |
| US7057026B2 (en) | 2001-12-04 | 2006-06-06 | Solexa Limited | Labelled nucleotides |
| US6815167B2 (en) | 2002-04-25 | 2004-11-09 | Geneohm Sciences | Amplification of DNA to produce single-stranded product of defined sequence and length |
| US20070065816A1 (en) | 2002-05-17 | 2007-03-22 | Affymetrix, Inc. | Methods for genotyping |
| US7108976B2 (en) | 2002-06-17 | 2006-09-19 | Affymetrix, Inc. | Complexity management of genomic DNA by locus specific amplification |
| WO2004001074A1 (en) * | 2002-06-21 | 2003-12-31 | Lynx Therapeutics, Inc. | Method for detecting foreign dna in a host genome |
| ES2550513T3 (es) | 2002-08-23 | 2015-11-10 | Illumina Cambridge Limited | Nucleótidos modificados para secuenciación de polinucleótidos |
| ES2694651T3 (es) | 2002-08-23 | 2018-12-26 | Illumina Cambridge Limited | Nucleótidos etiquetados |
| EP1546345B1 (en) | 2002-09-05 | 2007-03-28 | Plant Bioscience Limited | Genome partitioning |
| US20040157238A1 (en) | 2002-09-20 | 2004-08-12 | Quinn John J. | Method for detection of multiple nucleic acid sequence variations |
| EP1567669B1 (en) | 2002-12-02 | 2010-03-24 | Illumina Cambridge Limited | Determination of methylation of nucleic acid sequences |
| EP2361991A1 (en) | 2002-12-18 | 2011-08-31 | Third Wave Technologies, Inc. | Detection of small nucleic acids |
| JP2004208586A (ja) | 2002-12-27 | 2004-07-29 | Wakunaga Pharmaceut Co Ltd | Hlaの検出 |
| US7970548B2 (en) | 2003-01-10 | 2011-06-28 | Keygene N.V. | AFLP-based method for integrating physical and genetic maps |
| EP1594981B1 (en) * | 2003-01-29 | 2010-03-17 | 454 Corporation | Double ended sequencing |
| GB0304371D0 (en) | 2003-02-26 | 2003-04-02 | Solexa Ltd | DNA Sequence analysis |
| CA2555962C (en) | 2003-02-26 | 2015-10-06 | Callida Genomics, Inc. | Random array dna analysis by hybridization |
| JP4888876B2 (ja) | 2003-06-13 | 2012-02-29 | 田平 武 | アルツハイマー病の治療のための組換えアデノ随伴ウィルスベクター |
| GB0320059D0 (en) | 2003-08-27 | 2003-10-01 | Solexa Ltd | A method of sequencing |
| WO2005026686A2 (en) | 2003-09-09 | 2005-03-24 | Compass Genetics, Llc | Multiplexed analytical platform |
| WO2005040400A2 (en) | 2003-10-24 | 2005-05-06 | Mmi Genomics, Inc. | Methods and systems for inferring traits to manage non-beef livestock |
| GB0326073D0 (en) | 2003-11-07 | 2003-12-10 | Solexa Ltd | Improvements in or relating to polynucleotide arrays |
| WO2005065814A1 (en) * | 2004-01-07 | 2005-07-21 | Solexa Limited | Modified molecular arrays |
| GB0400584D0 (en) | 2004-01-12 | 2004-02-11 | Solexa Ltd | Nucleic acid chacterisation |
| GB0400974D0 (en) | 2004-01-16 | 2004-02-18 | Solexa Ltd | Multiple inexact matching |
| US20050233354A1 (en) * | 2004-01-22 | 2005-10-20 | Affymetrix, Inc. | Genotyping degraded or mitochandrial DNA samples |
| GB0402895D0 (en) | 2004-02-10 | 2004-03-17 | Solexa Ltd | Arrayed polynucleotides |
| AU2005214329A1 (en) | 2004-02-12 | 2005-09-01 | Population Genetics Technologies Ltd | Genetic analysis by sequence-specific sorting |
| US7709262B2 (en) | 2004-02-18 | 2010-05-04 | Trustees Of Boston University | Method for detecting and quantifying rare mutations/polymorphisms |
| EP1574585A1 (en) | 2004-03-12 | 2005-09-14 | Plant Research International B.V. | Method for selective amplification of DNA fragments for genetic fingerprinting |
| US7220549B2 (en) | 2004-12-30 | 2007-05-22 | Helicos Biosciences Corporation | Stabilizing a nucleic acid for nucleic acid sequencing |
| US7407757B2 (en) | 2005-02-10 | 2008-08-05 | Population Genetics Technologies | Genetic analysis by sequence-specific sorting |
| US7393665B2 (en) | 2005-02-10 | 2008-07-01 | Population Genetics Technologies Ltd | Methods and compositions for tagging and identifying polynucleotides |
| WO2006122215A2 (en) | 2005-05-10 | 2006-11-16 | State Of Oregon Acting By & Through The State Board Of Higher Education On Behalf Of The University Of Oregon | Methods of mapping polymorphisms and polymorphism microarrays |
| JP2008546405A (ja) | 2005-06-23 | 2008-12-25 | キージーン ナムローゼ フェンノートシャップ | ハイスループットシーケンシング技術を使用して複雑なゲノムをシーケンシングするための改善された戦略 |
| EP2302070B1 (en) | 2005-06-23 | 2012-08-22 | Keygene N.V. | Strategies for high throughput identification and detection of polymorphisms |
| US20070020640A1 (en) | 2005-07-21 | 2007-01-25 | Mccloskey Megan L | Molecular encoding of nucleic acid templates for PCR and other forms of sequence analysis |
| CA2623539C (en) | 2005-09-29 | 2015-12-15 | Keygene N.V. | High throughput screening of mutagenized populations |
| WO2007055568A1 (en) | 2005-11-14 | 2007-05-18 | Keygene N.V. | Method for high throughput screening of transposon tagging populations and massive parallel sequence identification of insertion sites |
| US7537897B2 (en) | 2006-01-23 | 2009-05-26 | Population Genetics Technologies, Ltd. | Molecular counting |
-
2007
- 2007-04-04 DK DK07747276.9T patent/DK2002017T3/en active
- 2007-04-04 ES ES17163116T patent/ES2829295T3/es active Active
- 2007-04-04 PT PT151586211T patent/PT2963127T/pt unknown
- 2007-04-04 PL PL15158621T patent/PL2963127T3/pl unknown
- 2007-04-04 EP EP15158621.1A patent/EP2963127B1/en active Active
- 2007-04-04 EP EP07747276.9A patent/EP2002017B1/en active Active
- 2007-04-04 ES ES07747276.9T patent/ES2545264T3/es active Active
- 2007-04-04 JP JP2009504137A patent/JP5389638B2/ja active Active
- 2007-04-04 EP EP17163116.1A patent/EP3239304B1/en active Active
- 2007-04-04 DK DK17163116.1T patent/DK3239304T3/da active
- 2007-04-04 ES ES15158621.1T patent/ES2645661T3/es active Active
- 2007-04-04 WO PCT/NL2007/000094 patent/WO2007114693A2/en not_active Ceased
- 2007-04-04 US US12/296,009 patent/US20090253581A1/en not_active Abandoned
-
2012
- 2012-02-02 US US13/364,799 patent/US20120135871A1/en not_active Abandoned
- 2012-04-18 US US13/449,629 patent/US20120202698A1/en not_active Abandoned
-
2013
- 2013-07-29 JP JP2013156870A patent/JP2013215212A/ja not_active Withdrawn
-
2014
- 2014-05-22 US US14/285,430 patent/US10023907B2/en active Active
-
2018
- 2018-06-05 US US16/000,252 patent/US20180291439A1/en not_active Abandoned
-
2019
- 2019-07-19 US US16/516,866 patent/US20200181694A1/en not_active Abandoned
Also Published As
| Publication number | Publication date |
|---|---|
| US20180291439A1 (en) | 2018-10-11 |
| ES2545264T3 (es) | 2015-09-09 |
| US20120135871A1 (en) | 2012-05-31 |
| US20200181694A1 (en) | 2020-06-11 |
| EP3239304B1 (en) | 2020-08-19 |
| US20140303007A1 (en) | 2014-10-09 |
| DK2002017T3 (en) | 2015-09-07 |
| ES2645661T3 (es) | 2017-12-07 |
| JP5389638B2 (ja) | 2014-01-15 |
| EP3239304A1 (en) | 2017-11-01 |
| EP2963127A1 (en) | 2016-01-06 |
| US10023907B2 (en) | 2018-07-17 |
| US20090253581A1 (en) | 2009-10-08 |
| JP2013215212A (ja) | 2013-10-24 |
| WO2007114693A3 (en) | 2007-12-21 |
| PL2963127T3 (pl) | 2018-01-31 |
| HK1219761A1 (en) | 2017-04-13 |
| EP2963127B1 (en) | 2017-08-16 |
| EP2002017A2 (en) | 2008-12-17 |
| WO2007114693A2 (en) | 2007-10-11 |
| PT2963127T (pt) | 2017-10-06 |
| US20120202698A1 (en) | 2012-08-09 |
| EP2002017B1 (en) | 2015-06-10 |
| JP2009536817A (ja) | 2009-10-22 |
| HK1244301A1 (en) | 2018-08-03 |
| DK3239304T3 (da) | 2020-10-26 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| ES2829295T3 (es) | Detección de alto rendimiento de marcadores moleculares basados en AFLP y secuenciación de alto rendimiento | |
| ES2393318T3 (es) | Estrategias para la identificación y detección de alto rendimiento de polimorfismos | |
| ES2357549T3 (es) | Estrategias para la identificación y detección de alto rendimiento de polimorfismos. | |
| ES2338459T5 (es) | Exploración de alto rendimiento de poblaciones mutagenizadas | |
| ES2882401T3 (es) | Método de detección de polimorfismos basado en AFLP de alto rendimiento | |
| US20130331277A1 (en) | Paired end random sequence based genotyping | |
| CN101432438A (zh) | 基于aflp和高通量测序的对分子标记的高通量检测 | |
| WO2024033411A1 (en) | Methods for determining the location of a target sequence and uses | |
| HK1244301B (en) | High throughput detection of molecular markers based on aflp and high troughput sequencing | |
| HK1219761B (en) | High throughput detection of molecular markers based on restriction fragments |