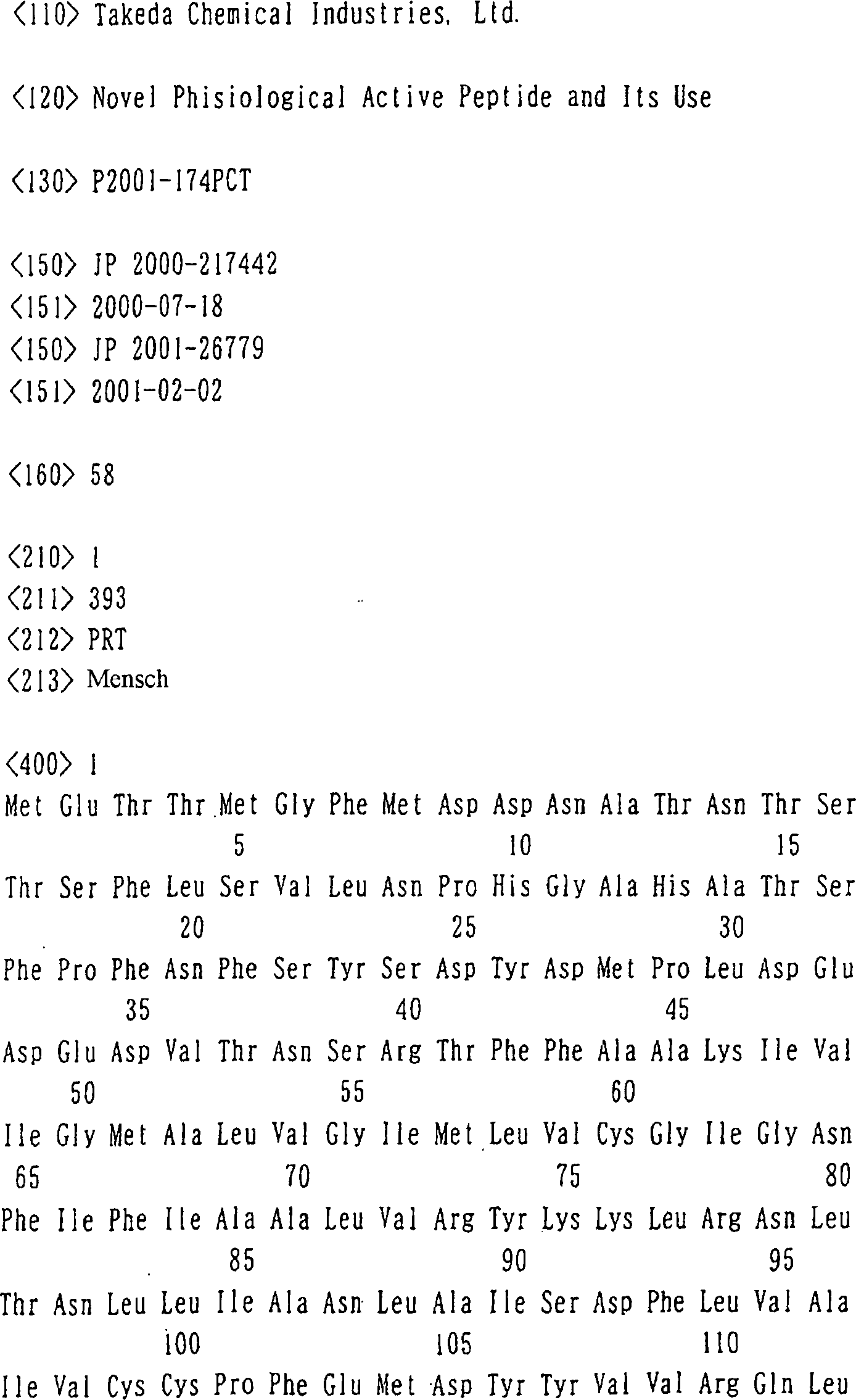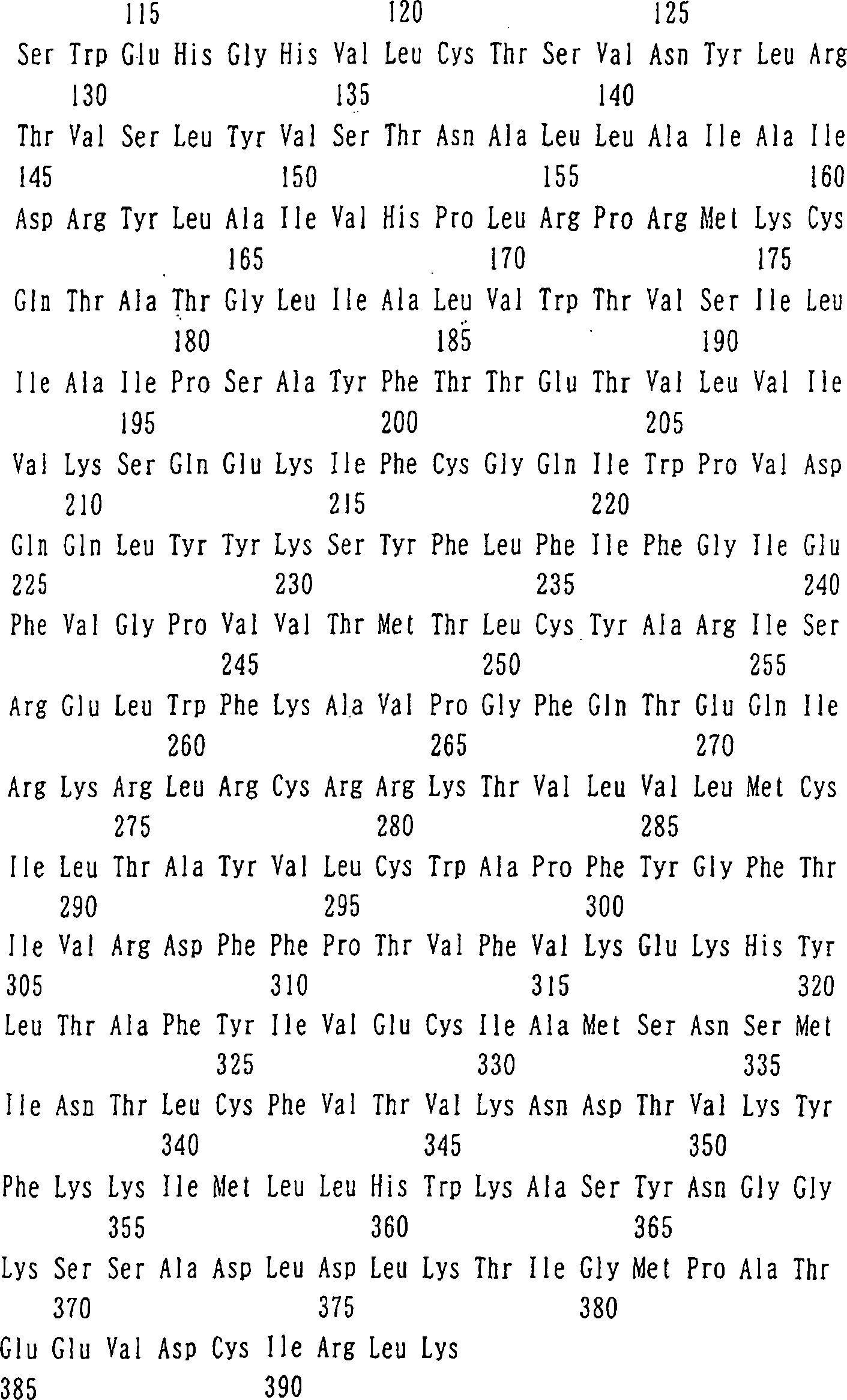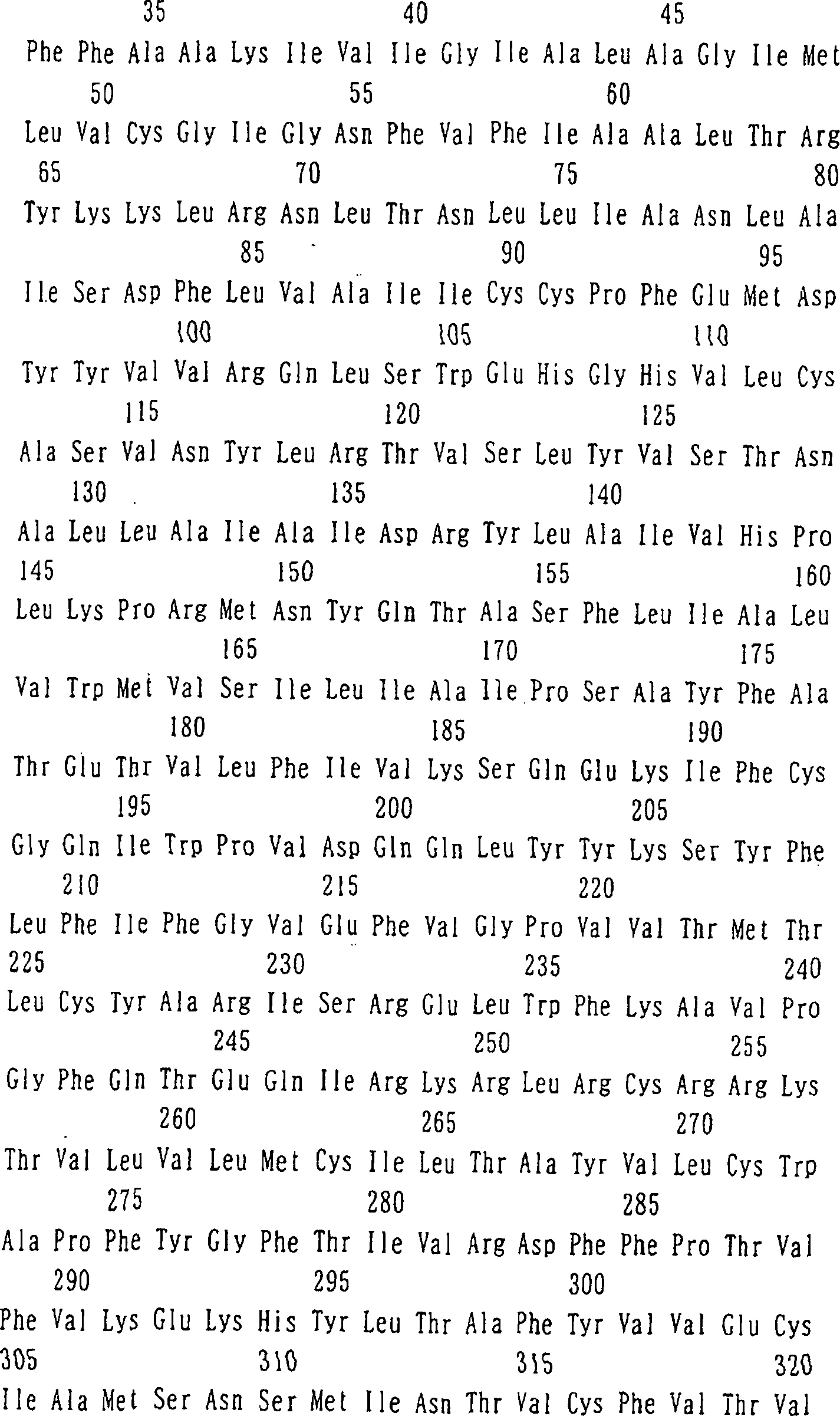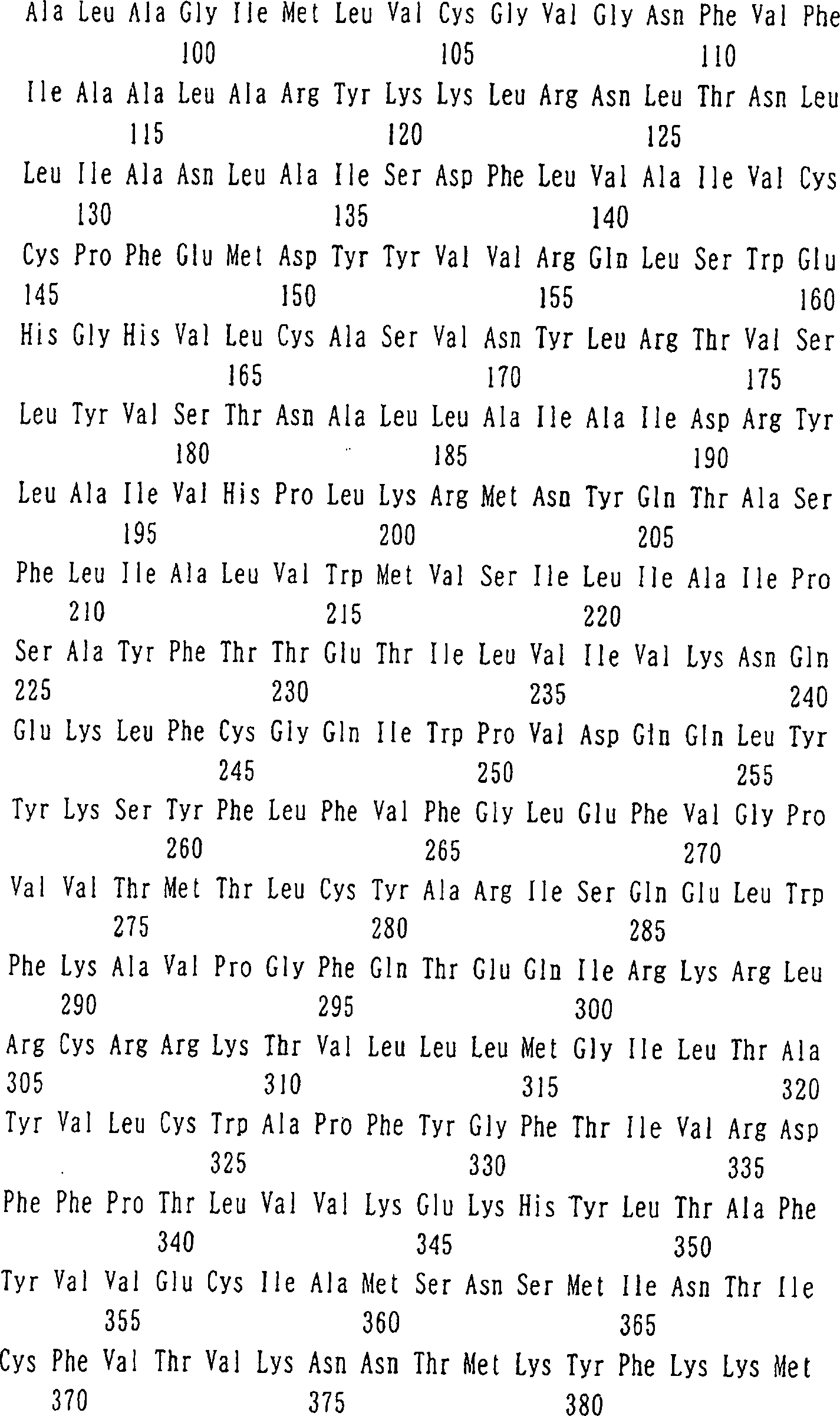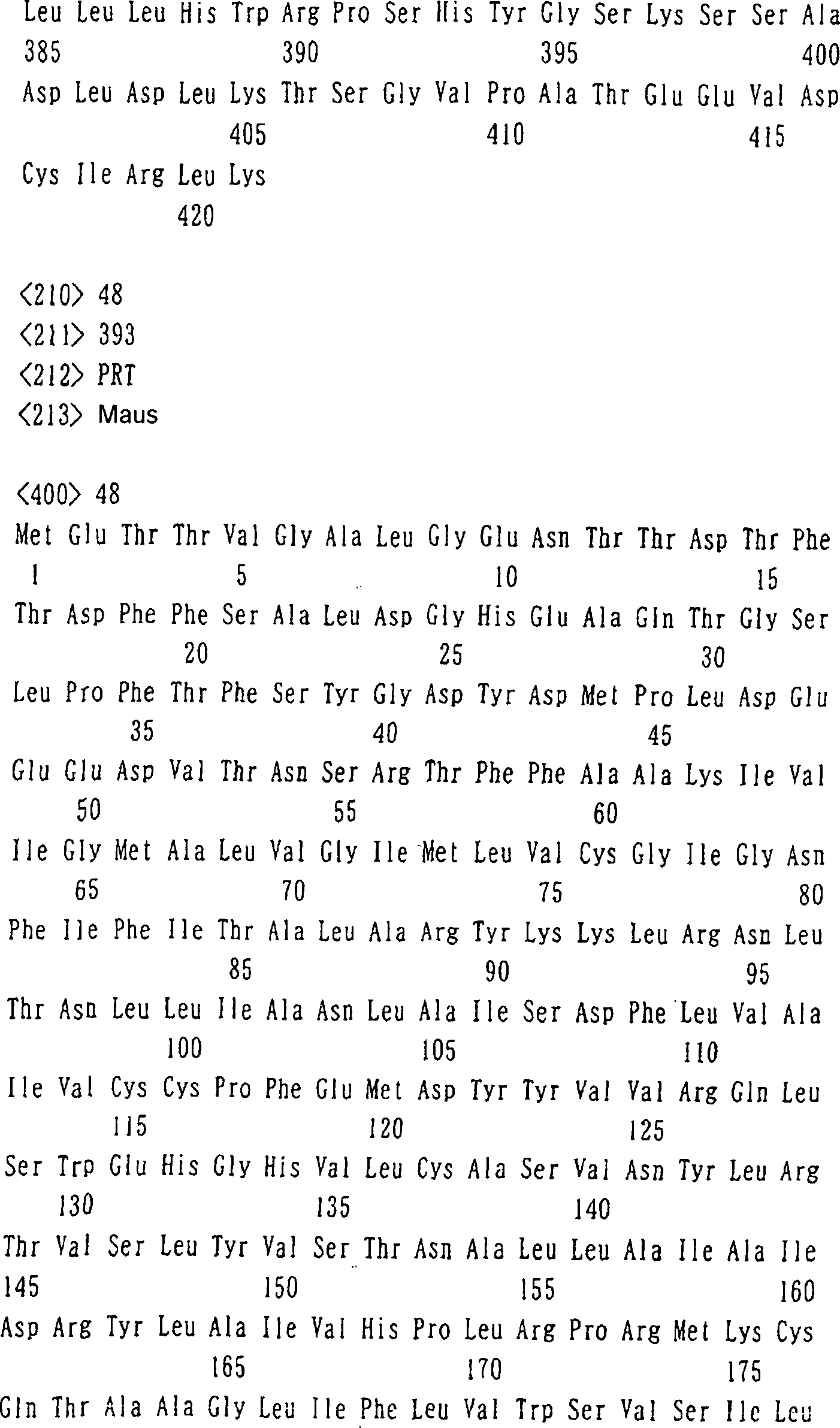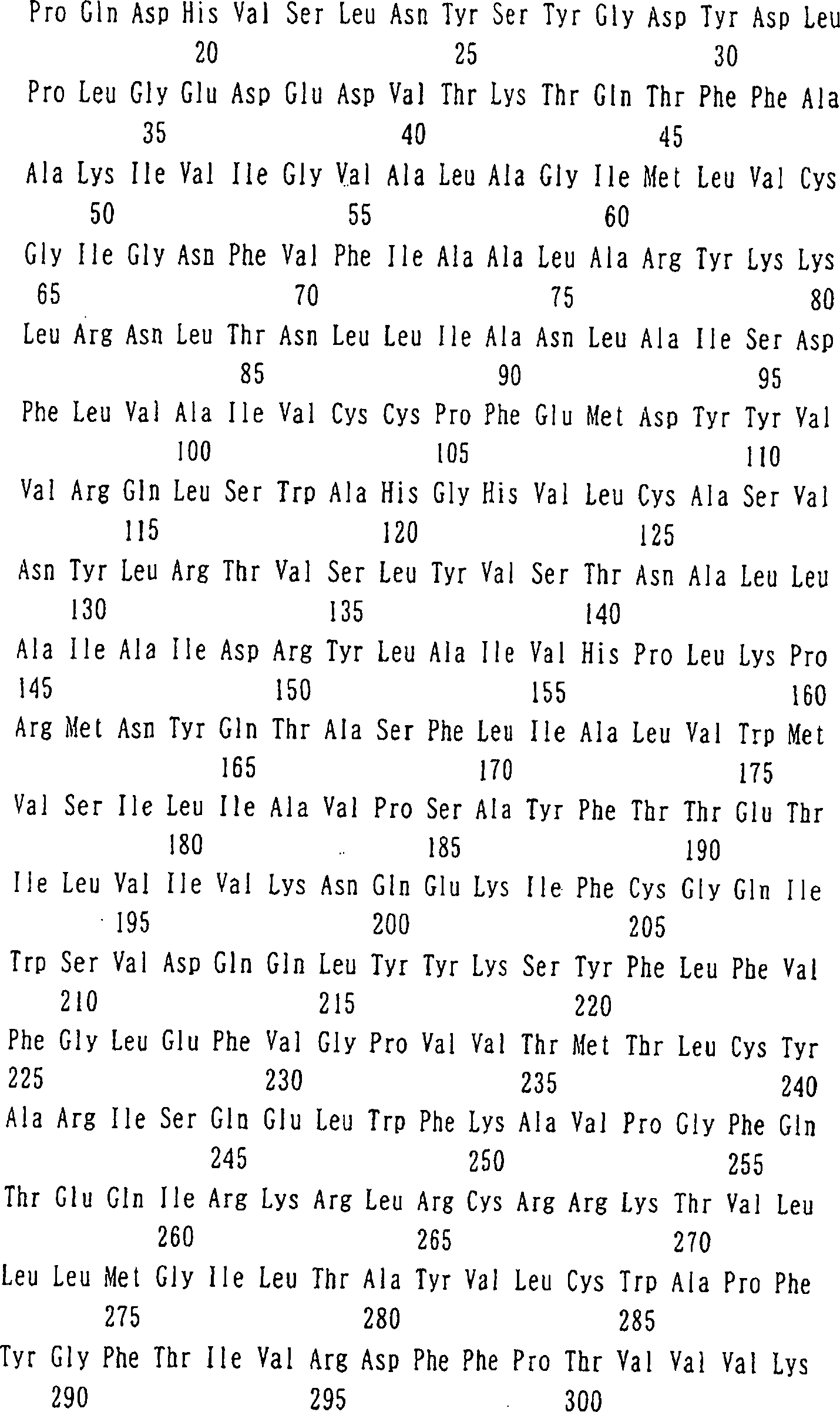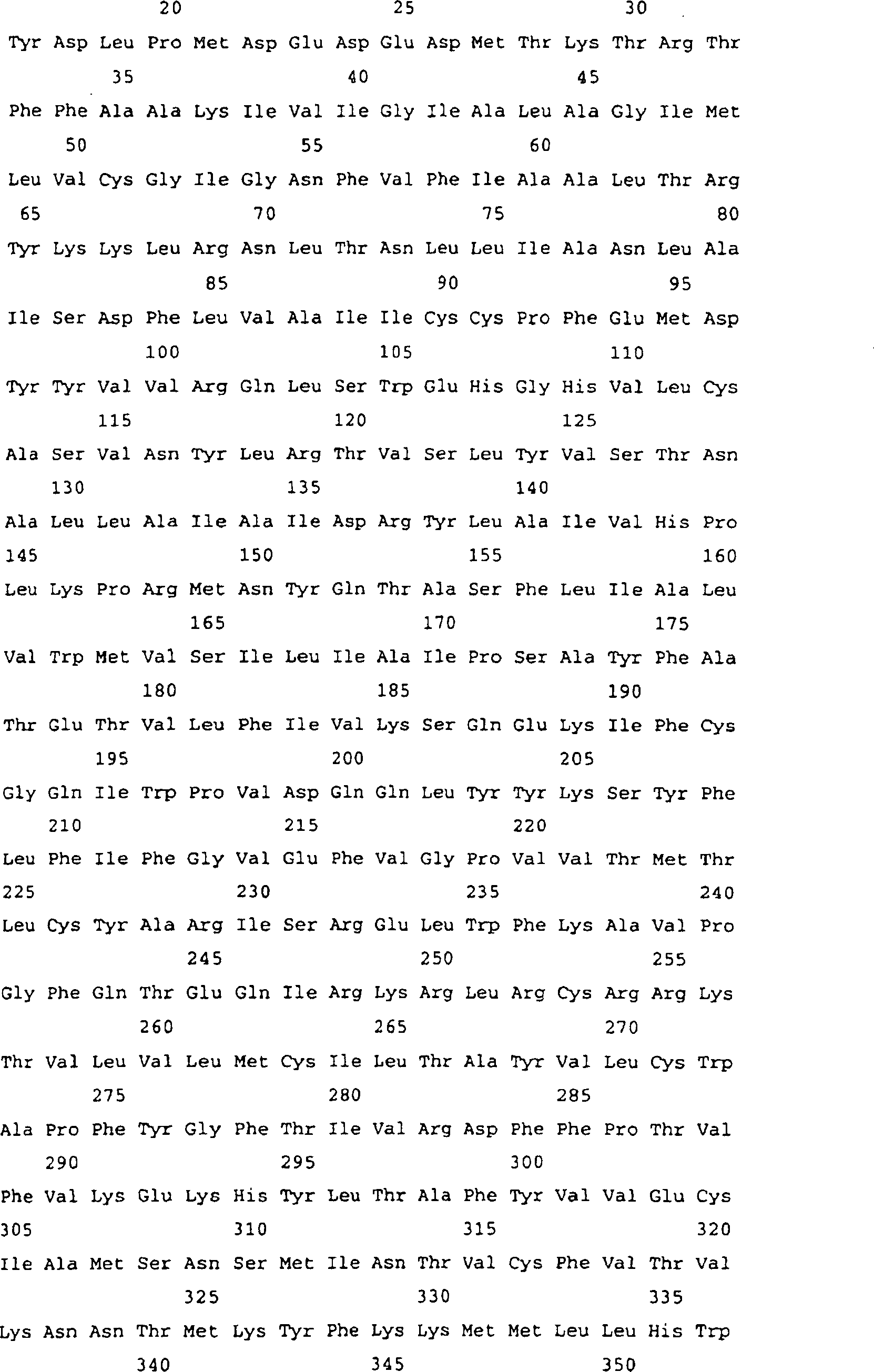-
GEBIET DER
ERFINDUNG
-
Die
vorliegende Erfindung bezieht sich auf (1) ein Peptid, dadurch gekennzeichnet,
das es dieselbe oder im wesentlichen dieselbe Aminosäuresequenz
wie die Aminosäuresequenz
enthält,
die durch SEQ ID NR. 20 oder SEQ ID NR. 21 dargestellt ist, oder
ein Salz davon.
-
STAND DER
TECHNIK
-
Wichtige
biologische Funktionen, einschließlich Aufrechterhaltung der
Homöostasie
in dem lebenden Körper,
Reproduktion, Entwicklung von Individuen, Metabolismus, Wachstum,
Kontrolle des Nerven-, Zirkulations-, Immun-, Verdauungs- und Stoffwechselsystems,
sensorische Anpassung usw., werden durch Zellen reguliert, die endogene
Faktoren empfangen, wie verschiedene Hormone und Neurotransmitter,
oder Sinnesstimulation, wie Licht oder Geruch, über spezifische Rezeptoren,
die auf Zellmembranen vorliegen, die für diese Faktoren oder Stimulation
reserviert sind und mit ihnen interagieren. Viele von diesen Rezeptoren
für Hormone oder
Neurotransmitter werden durch diese funktionelle Regulierung an
Guanin-Nucleotid-bindende
Proteine gekoppelt (hierin nachstehend manchmal nur als G-Proteine
bezeichnet), und sind durch Entwickeln einer Vielzahl von Funktionen
durch intrazelluläre
Signaltransduktion über
die Aktivierung der G-Proteine gekennzeichnet. Außerdem verfügen diese
Rezeptorproteine über
sieben gemeinsame Transmembrandomänen. Aus den obigen Gründen werden
diese Rezeptoren daher gemeinsam als G-Protein-gekoppelte Rezeptoren
oder sieben-Transmembranrezeptoren bezeichnet. Wie oben angegeben,
ist es bekannt, daß verschiedene
Hormone oder Neurotransmitter und ihre Rezeptorproteine vorliegen
und miteinander interagieren, um wichtige Rollen bei der Regulierung
der biologischen Funktionen zu spielen. Jedoch ist es noch schlecht
verstanden, ob irgendwelche anderen unbekannten Substanzen (Hormone,
Neurotransmitter usw.) und Rezeptoren für diese Substanzen vorliegen.
-
In
jüngsten
Jahren ist das menschliche Gen bei einem zunehmenden Tempo durch
gesammelte Sequenzinformationen durch Sequenzieren der menschlichen
genomischen DNA oder verschiedener menschlicher Gewebe-abgeleiteter
cDNA bei zufälligem
und schnellem Fortschritt in der Genanalysetechnologie aufgeklärt worden.
Basierend auf dem Vorhergehenden wird manifestiert, daß es viele
Gene gibt, von denen angenommen wird, daß sie Proteine mit unbekannten
Funktionen kodieren. G-Protein-gekoppelte Rezeptoren haben nicht
nur sieben Transmembrandomänen,
sondern viele gemeinsame Sequenzen liegen in ihren Nukleinsäuren oder
Aminosäuren
vor, und können
daher klar als G-Protein-gekoppelte Rezeptoren in diesen Proteinen
identifiziert werden. Andererseits werden diese G-Protein-gekoppelten
Rezeptorgene durch Polymerase-Kettenreaktion (hierin nachstehend
als PCR abgekürzt)
unter Verwendung einer solchen Ähnlichkeit
in der Struktur erhalten (Nature Cell Biology, 2, 703–708 (2000)).
Bei diesen bis jetzt so erhaltenen G-Protein-gekoppelten Rezeptoren
können
Liganden für
einige Rezeptoren, die Subtypen mit hoher Homologie in der Struktur für bekannte
Rezeptoren sind, ohne weiteres vorhersehbar sein, aber in den meisten
Fällen
sind ihre endogenen Liganden unvorhersehbar, so daß keine
Liganden, die diesen Rezeptoren entsprechen, gefunden werden. Aus
diesem Grund werden diese Rezeptoren Orphan-Rezeptoren genannt.
Es ist wahrscheinlich, daß nicht identifizierte
endogene Liganden für
diese Orphan-Rezeptoren
an dem schlecht analysierten biologischen Phänomen beteiligt sein würden, da
die Liganden unbekannt waren. Und wenn diese Liganden mit wichtigen physiologischen
Wirkungen oder pathologischen Zuständen in Verbindung gebracht
werden, wird erwartet, daß die
Entwicklung dieser Rezeptoragonisten und -antagonisten zum Durchbruch
von neuen Arzneimitteln führen
wird (Stadel, J. et al., TiPS, 18, 430–437, 1997; Marchese, A. et
al., TiPS, 20, 370–375,
1999; Civelli, O. et al., Brain Res., 848, 63–65, 1999). Bis jetzt gibt
es jedoch wenige Beispiele, um Liganden für Orphan-G-Protein-gekoppelte
Rezeptoren tatsächlich
zu identifizieren.
-
Kürzlich versuchten
einige Gruppen, Liganden für
diese Orphan-Rezeptoren zu identifizieren, und berichteten über die
Isolation von Liganden, die neue physiologisch wirksame Peptide
sind, und die Bestimmung ihrer Strukturen. Reinsheid et al. und
Meunier et al. führten
unabhängig
voneinander cDNA, die Orphan-G-Protein-gekoppelten Rezeptor LC132
oder ORL1 codiert, in Tierzellen ein, um einen Rezeptor zu exprimieren,
isolierten ein neues Peptid aus Schweinehirn- und Rattenhirnextrakt
unter Verwendung der Antwort des Rezeptors als ein Indikator, der
in bezug auf seine Antwort Orphanin FQ oder Nociceptin genannt wird,
und bestimmten seine Sequenz (Reinsheid, R. K. et al., Science,
270, 792–794,
1995; Meunier, J.-C. et al., Nature, 377, 532–535, 1995). Es wurde berichtet,
daß dieses
Peptid mit einem Schmerzgefühl
verbunden ist. Weitere Untersuchungen an dem Rezeptor in der Knockout-Maus offenbarten,
daß das
Peptid im Gedächtnis
involviert war (Manabe, T. et al., Nature, 394, 577–581, 1998).
-
Folglich
wurden neue Peptide, einschließlich
PrRP (Prolactin-releasing Peptid), Orexin, Apelin, Ghrelin und GALP
(Galanin-artiges Peptid) usw. als Liganden für Orphan-G-Protein-gekoppelte Rezeptoren
isoliert (Hinuma, S. et al., Nature, 393, 272–276, 1998; Sakurai, T. et
al., Cell, 92, 573–585,
1998; Tatemoto, K. et al., Bichem. Biophys. Res. Commun., 251, 471–476, 1998;
Kojima, M. et al., Nature, 402, 656–660, 1999; Ohtaki, T. et al.,
J. Biol. Chem., 274, 37041–37045,
1999).
-
Andererseits
werden einige Rezeptoren für
physiologisch aktive Peptide, die bisher unbekannt sind, durch ähnliche
Verfahren aufgedeckt. Es wurde aufgedeckt, daß ein Rezeptor für Motilin,
der mit der Kontraktion der Darmtrakte verbunden ist, GPR38 war
(Feighner, S. D. et al., Science, 284, 2184–2188, 1999). Außerdem wurde
SLC-1 als ein Rezeptor für
das Melanin-konzentrierende Hormon (MCH) identifiziert (Chambers, J.
et al., Nature, 400, 261–265,
1999; Saito, Y. et al., Nature, 400, 265–269, 1999; Shimomura, Y. et
al., Biochem. Biophys. Res. Commun., 261, 622–626, 1999; Lembo, P. M. C.
et al., Nature Cell Biol., 1, 267–271, 1999; Bachner, D. et
al., FEBS Lett., 457, 522–524,
1999). Ebenso wurde berichtet, daß GPR14 (SENR) ein Rezeptor
für Urotensin
II ist (Ames, R. S. et al., Nature, 401, 282–286, 1999; Mori, M. et al.,
Biochem. Biophys. Res. Commun., 265, 123–129, 1999; Nothakker, H.-P.
et al., Nature Cell Biol., 1, 383–385, 1999; Liu, Q. et al., Biochem.
Biophys. Res. Commun., 266, 174–178,
1999). Es wurde gezeigt, daß MCH
an Fettleibigkeit beteiligt ist, da ihre Knockout-Mäuse einen
hypophagischen und mageren Phänotyp
zeigten (Shimada, M. et al., Nature, 396, 670–674, 1998), und da ihr Rezeptor
aufgedeckt wurde, wurde es möglich,
einen Rezeptorantagonisten zu untersuchen, der wahrscheinlich als
ein Antifettsuchtmittel nützlich
sein sollte. Es wurde außerdem berichtet,
daß Urotensin
II eine starke Wirkung auf das Herz-Kreislauf System zeigt, da es
Herzischämie
durch intravenöse
Injektion an einen Affen induziert (Ames, R. S. et al., Nature,
401, 282–286,
1999).
-
Wie
oben beschrieben, sind Orphan-Rezeptoren und Liganden dafür oftmals
an einer neuen physiologischen Aktivität beteiligt, deren Erforschung
zur Entwicklung von neuen Arzneimitteln führen wird. Jedoch sind die
Untersuchungen der Liganden für
Orphan-Rezeptoren von vielen Schwierigkeiten begleitet. Während die
Gegenwart von vielen Orphan-Rezeptoren bisher aufgedeckt wurde,
wurden nur wenige Liganden für
diese Rezeptoren entdeckt.
-
Die
betreffenden Erfinder fanden einen neuen Rezeptor ZAQ, ebenso offenbart
in WO 0034334 und WO 9846620, der ein Orphan-G-Protein-gekoppelter
Rezeptor ist (ein Protein, enthaltend dieselbe oder im wesentlichen
dieselbe Aminosäuresequenz
wie die Aminosäuresequenz,
die durch SEQ ID NR. 1 der Beschreibung dargestellt ist: hierin
nachstehend manchmal einfach als ZAQ in der Beschreibung bezeichnet).
Jedoch war es bis jetzt unbekannt, was der Ligand war.
-
Es
ist das Problem gewesen, einen Liganden für das Orphan-Rezeptorprotein
ZAQ zu finden und ein Verfahren zum Screenen einer Verbindung zu
etablieren, gekennzeichnet durch die Verwendung von ZAQ und einem
Liganden dafür.
-
OFFENBARUNG DER ERFINDUNG
-
Die
betreffenden Erfinder fanden eine Substanz mit einer Ligandenaktivität, die für ZAQ spezifisch
ist, die in einem Milchextrakt vorlag, isolierten die Substanz und
bestimmten ihre Struktur. Die Erfinder fanden ebenso ein Gen, das
ein humanes Peptid von dieser aktiven Komponente kodiert, versuchten,
das Gen in Tierzellen zu exprimieren, und bestätigten, daß eine peptidähnliche
Substanz, die die ZAQ-exprimierten Zellen aktivieren kann, in dem
Kulturüberstand
sekretiert wurde.
-
Basierend
auf diesen Ergebnissen fanden die betreffenden Erfinder heraus,
daß Arzneimittel
für die Behandlung
von Krankheiten, die durch ZAQ vermittelt werden (ZAQ-Antagonisten
oder -agonisten usw., speziell Mittel zur Vorbeugung/Behandlung
von Verdauungskrankheiten usw.) durch das Screeningsystem unter Verwendung
von ZAQ und einem ZAQ-Ligandenpeptid
gescreent werden können.
-
Das
heißt,
die vorliegende Erfindung bezieht sich auf folgende Merkmale:
- (1) Peptid, bestehend aus der Aminosäuresequenz,
die in SEQ ID NR. 20 oder SEQ ID NR. 21 dargestellt ist, oder ein
Salz davon;
- (2) Peptid oder ein Salz gemäß (1), das
aus der in SEQ ID NR. 20 dargestellten Aminosäuresequenz besteht;
- (3) Peptid oder ein Salz gemäß (l), das
aus der in SEQ ID NR. 21 dargestellten Aminosäuresequenz besteht;
- (4) Polynukleotid, bestehend aus einem Polynukleotid, das für das Peptid
gemäß (1) kodiert;
- (5) Polynukleotid gemäß (4), das
eine DNA ist;
- (6) DNA gemäß (5), enthaltend
die Basensequenz, die in SEQ ID NR. 26 oder SEQ ID NR. 27 dargestellt
ist;
- (7) Rekombinanter Vektor, enthaltend das Polynukleotid gemäß (4);
- (8) Transformant, transformiert mit dem rekombinanten Vektor
gemäß (7);
- (9) Verfahren zur Herstellung des Peptids oder seines Salzes
gemäß (1), das
umfaßt,
daß man
den Transformanten von (8) kultiviert und das Peptid gemäß (1) produziert/akkumuliert.
- (10) Antikörper
gegen das Peptid oder sein Salz gemäß (1).
-
AUSFÜHRLICHE
BESCHREIBUNG DER ZEICHNUNGEN
-
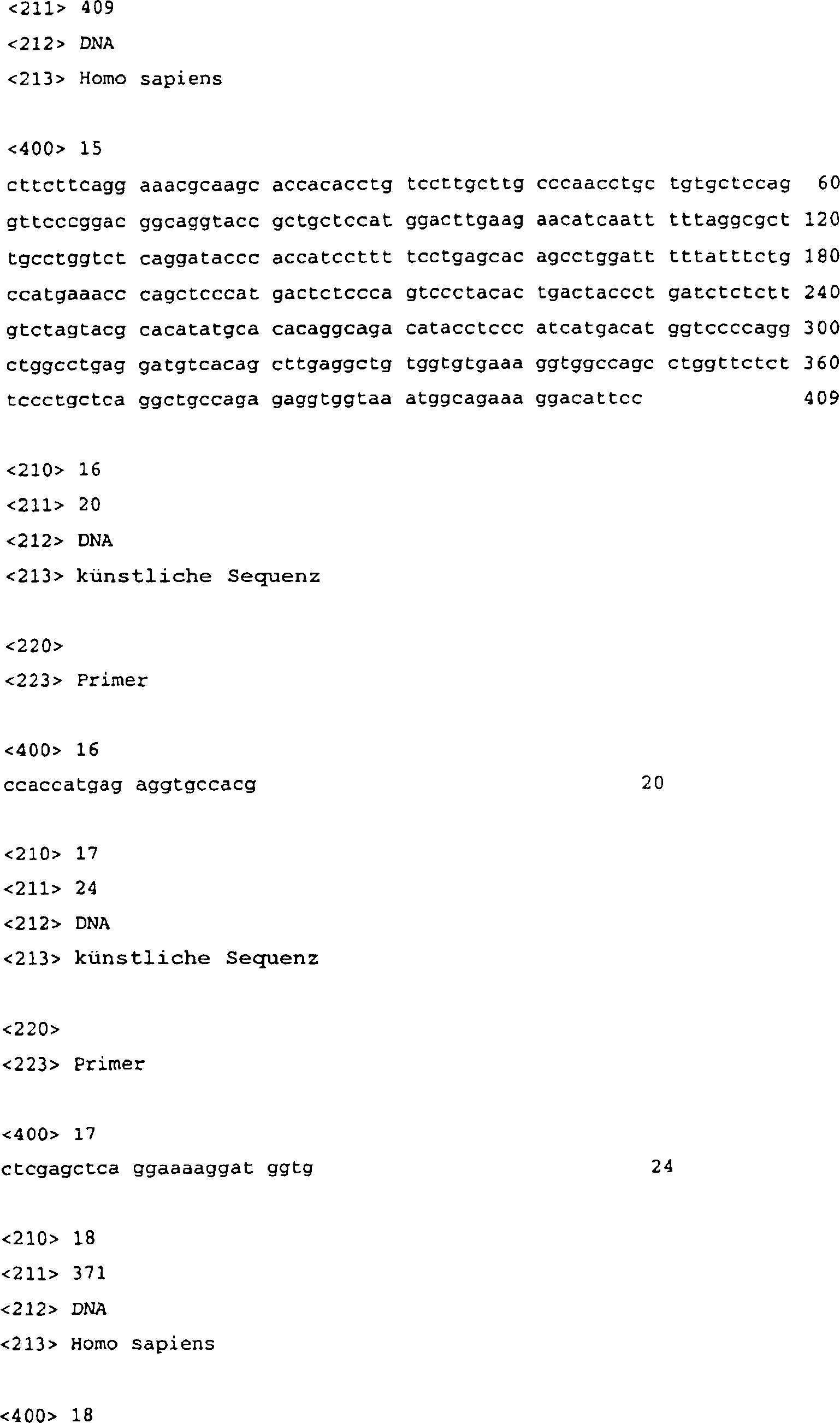
1 zeigt
die Basensequenz der DNA, die das menschliche Gehirn-abgeleitete
Protein der Erfindung kodiert, erhalten in BEISPIEL 1 (ZAQC), und
die daraus abgeleitete Aminosäuresequenz
(fortgesetzt auf 2).
-
2 zeigt
die Basensequenz der DNA, die das menschliche Gehirn-abgeleitete
Protein der Erfindung kodiert, erhalten in BEISPIEL 1 (ZAQC), und
die daraus abgeleitete Aminosäuresequenz
(fortgesetzt von 2 bis 3).
-
3 zeigt
die Basensequenz der DNA, die das menschliche Gehirn-abgeleitete
Protein der Erfindung kodiert, erhalten in BEISPIEL 1 (ZAQC), und
die daraus abgeleitete Aminosäuresequenz
(fortgesetzt von 2).
-
4 zeigt
die Basensequenz der DNA, die das menschliche Gehirn-abgeleitete
Protein der Erfindung kodiert, erhalten in BEISPIEL 1 (ZAQT), und
die daraus abgeleitete Aminosäuresequenz
(fortgesetzt auf 5).
-
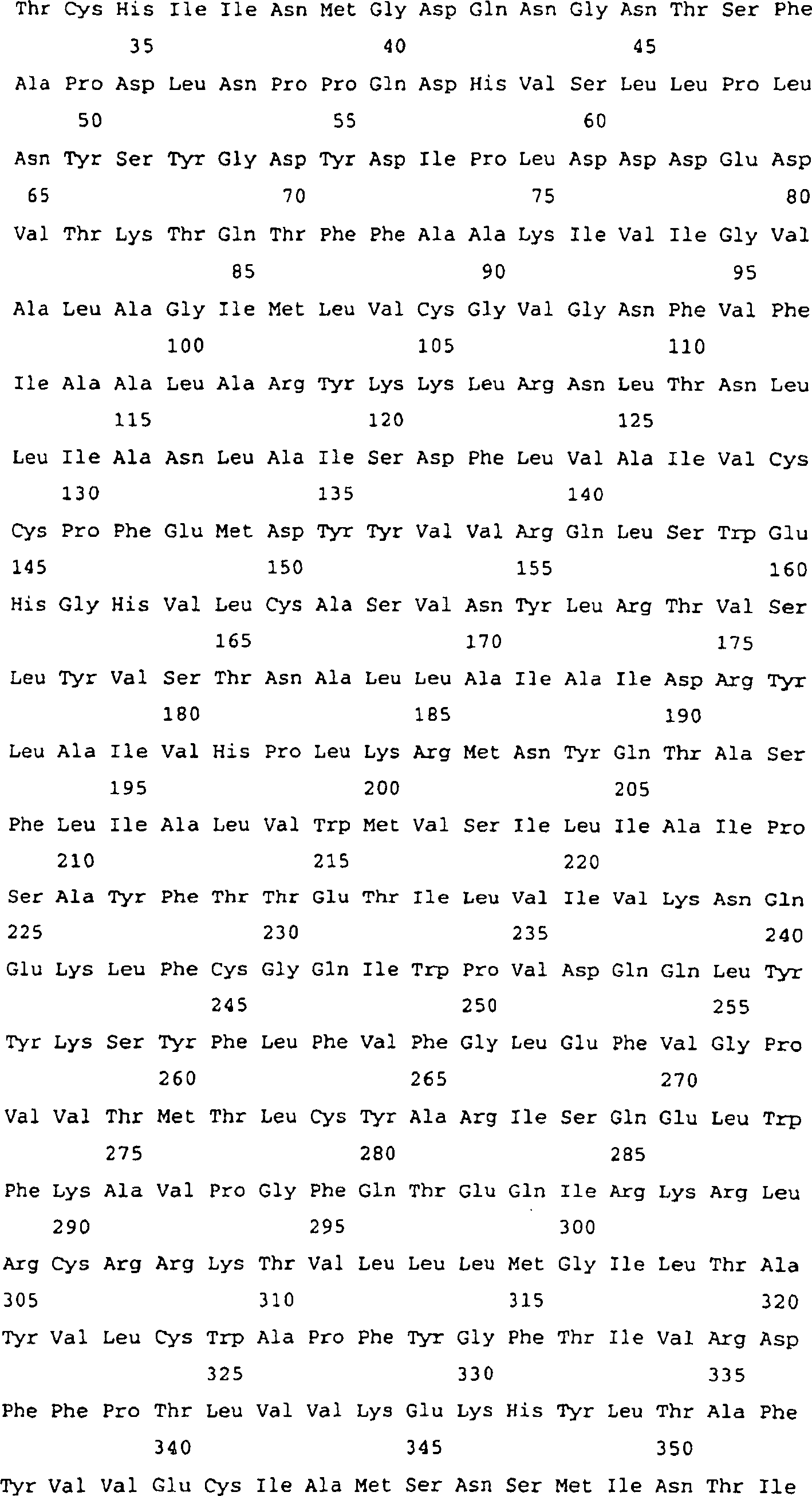
5 zeigt
die Basensequenz der DNA, die das menschliche Gehirn-abgeleitete
Protein der Erfindung kodiert, erhalten in BEISPIEL 1 (ZAQT), und
die daraus abgeleitete Aminosäuresequenz
(fortgesetzt von 4 bis 6).
-
6 zeigt
die Basensequenz der DNA, die das menschliche Gehirn-abgeleitete
Protein der Erfindung kodiert, erhalten in BEISPIEL 1 (ZAQT), und
die daraus abgeleitete Aminosäuresequenz
(fortgesetzt von 5).
-
7 zeigt
ein Diagramm der Hydrophobie für
das menschliche Gehirn-abgeleitete Protein der Erfindung.
-
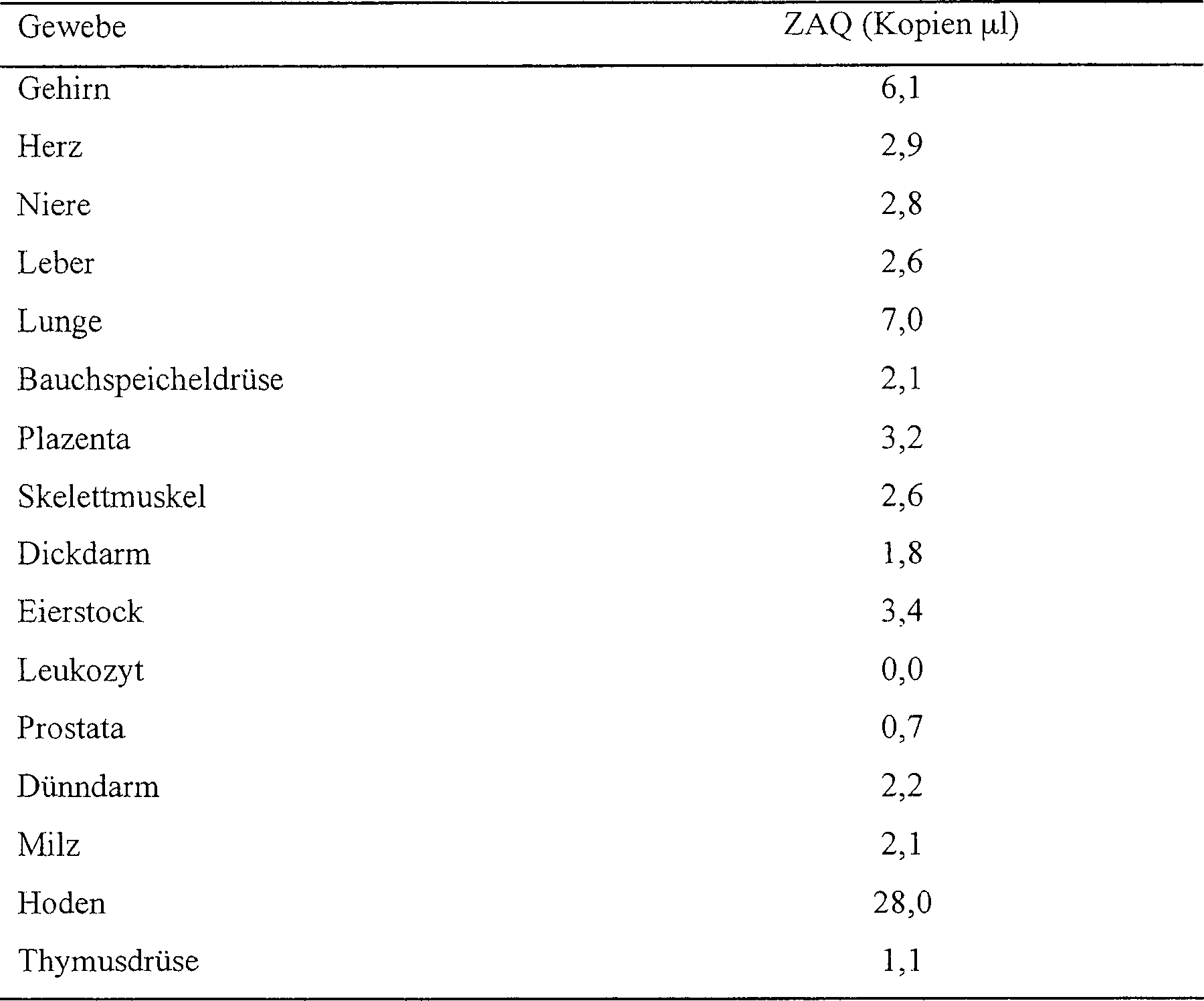
8 zeigt
die Ergebnisse der Expressions-Verteilungsanalyse von ZAQ, die in
BEISPIEL 2 durchgeführt
wurde.
-
9 zeigt
die Aminosäuresequenzen
von MIT1, menschentypischem ZAQ-Liganden-Präkursorpeptid
(A-Typ) und menschentypischem ZAQ-Liganden-Präkursorpeptid (G-Typ), wobei „MIT1," „Mensch (A-Typ)" und „Mensch
(G-Typ)" die Aminosäuresequenzen
von MIT1, menschentypischem reifem ZAQ-Ligandenpeptid (A-Typ) bzw.
menschentypischem reifem ZAQ-Ligandenpeptid (G-Typ) angibt.
-
10 zeigt die Ergebnisse der Analyse für die ZAQ-aktivierende
Wirkung durch das ZAQ-Ligandenpeptid,
gereinigt in BEISPIEL 6 (6-3).
-
11 zeigt eine Restriktionsenzymkarte von Plasmid
pCAN618, verwendet in BEISPIEL 5 (5-11).
-
12 zeigt die Ergebnisse der Messung für die ZAQ-Rezeptor-aktivierenden
Wirkungen durch menschentypisches ZAQ-Ligandenpeptid und MIT1, analysiert
in BEISPIEL 8 (8-3), wobei -o- und -•- menschentypisches ZAQ-Ligandenpeptid
bzw. MIT1 darstellen.
-
13 zeigt die Ergebnisse der Messung für I5E-Rezeptor-aktivierende
Wirkungen durch menschentypisches ZAQ-Ligandenpeptid und MIT1, analysiert
in BEISPIEL 8 (8-3), wobei -o- und -•- menschentypisches ZAQ-Ligandenpeptid
bzw. MIT1 darstellen.
-
14 zeigt die Ergebnisse der Messung für die kontraktile
Aktivität,
analysiert in BEISPIEL 11, wobei -∎- und -•- menschentypisches
ZAQ-Ligandenpeptid bzw. MIT1 darstellen.
-
15 zeigt die Ergebnisse der Messung für den Bindungsassay,
durchgeführt
in BEISPIEL 12 unter Verwendung der Membranfraktion von ZAQ, wobei
-∎- die 125I-MIT1-spezifische Bindungsmenge
darstellt, wenn menschentypisches ZAQ-Ligandenpeptid in variablen
Konzentrationen (wie auf der x-Achse angegeben) als eine Testverbindung
zugegeben wurde, und -•-
die 125I-MIT1-spezifische Bindungsmenge
angibt, wenn MIT1 ebenso als eine Testverbindung zugegeben wurde.
-
16 zeigt die Ergebnisse der Messung für den Bindungsassay,
durchgeführt
in BEISPIEL 12 unter Verwendung der Membranfraktion von I5E, wobei
-☐- die 125I-MIT1-spezifische Bindungsmenge
darstellt, wenn menschentypisches ZAQ-Ligandenpeptid in variablen
Konzentrationen (wie auf der x-Achse angegeben) als eine Testverbindung
zugegeben wurde, und -o- die 125I-MIT1-spezifische
Bindungsmenge darstellt, wenn MIT1 in variablen Konzentrationen
(wie auf der x-Achse angegeben) als eine Testverbindung zugegeben
wurde.
-
BESTE WEISE
ZUR DURCHFÜHRUNG
DER ERFINDUNG
-
Das
erfindungsgemäße Peptid
oder sein Salz ist ein Peptid oder sein Salz, das an ein Protein,
das dieselbe oder im wesentlichen dieselbe Aminosäuresequenz
wie die Aminosäuresequenz
enthält,
die durch SEQ ID NR. 1 dargestellt ist, oder ein Salz davon binden
kann (hierin nachstehend manchmal einfach als das gegenwärtige Protein
bezeichnet), und ist ein Peptid mit der Fähigkeit zum Binden an das gegenwärtige Protein,
um selbiges zu aktivieren, oder ein Salz davon.
-
Außerdem ist
das erfindungsgemäße Peptid
oder sein Salz ein Peptid oder sein Salz, das an ein Protein, das
dieselbe oder im wesentlichen dieselbe Aminosäuresequenz wie die Aminosäuresequenz
enthält,
die durch SEQ ID NR. 36, 40, 47, 48 oder 49 dargestellt ist, oder
ein Salz davon binden kann, und ist ein Peptid mit der Fähigkeit
zum Binden an das Protein der Erfindung, um selbiges zu aktivieren,
oder ein Salz davon.
-
Das
erfindungsgemäße Peptid
oder sein Salz ist ein Peptid, bestehend aus der durch SEQ ID NR.
20 oder SEQ ID NR. 21 dargestellten Aminosäuresequenz, und mit der Fähigkeit
zum Binden an das gegenwärtige
Protein der Erfindung, um selbiges zu aktivieren, oder ein Salz
davon.
-
Die
Fähigkeit
des erfindungsgemäßen Peptids
oder seines Salzes, an das Protein der Erfindung zu binden und das
Protein der Erfindung zu aktivieren, kann durch das später beschriebene
V erfahren analysiert werden.
-
Hierin
nachstehend wird das Peptid oder sein Salz der Erfindung manchmal
nur als das erfindungsgemäße Peptid
bezeichnet.
-
Das
gegenwärtige
Protein (G-Protein-gekoppeltes Rezeptorprotein) ist ein Rezeptorprotein,
enthaltend dieselbe oder im wesentlichen dieselbe Aminosäuresequenz
wie die Aminosäuresequenz
(die Aminosäuresequenz
in 1 bis 3 oder 4 bis 6),
die durch SEQ ID NR. 1 dargestellt ist.
-
Das
erfindungsgemäße Peptid
und das gegenwärtige
Protein der Erfindung kann irgendein Peptid oder Protein sein, das
von jeglichen Zellen von Menschen oder anderen Säugern abgeleitet wird (beispielsweise
Meerschweinchen, Ratte, Maus, Kaninchen, Schwein, Schaf Rind, Affe
usw.) (beispielsweise Milzzelle, Nervenzelle, Gliazelle, β-Zelle der
Bauchspeicheldrüse,
Knochenmarkszelle, Mesangiumzelle, Langerhans-Zelle, Epidermiszelle,
Epithelzelle, Endothelzelle, Fibroblast, Fibrozyt, Myozyt, Fettzelle,
Immunzelle (beispielsweise Makrophage, T-Zelle, B-Zelle, natürliche Killerzelle,
Mastzelle, Neutrophile, Basophile, Eosinophile, Monozyt), Megakaryozyt,
Synoviazelle, Knorpelzelle, Knochenzelle, Osteoblast, Osteoklast,
Brustdrüsenzelle,
Hepatozyt, Interstitialzelle usw., die entsprechenden Präkursorzellen,
Stammzellen, Krebszellen usw.) und Hämozytzellen (beispielsweise
MEL, M1, CTLL-2, HT-2, WEHI-3, HL-60, JOSK-1, K562, ML-1, MOLT-3,
MOLT-4, MOLT-10, CCRF-CEM, TALL-1, Jurkat, CCRT-HSB-2, KE-37, SKW-3,
HUT-78, HUT-102, H9, U937, THP-1, HEL, JK-1, CMK, KO-812, MEG-01
usw.); oder jegliche Gewebe, in denen diese Zellen vorliegen, wie
Gehirn oder jegliche Gehirnregionen (beispielsweise Riechkolben,
Mandelkern, zerebraler Basalbulbus, Hippocampus, Thalamus, Hypothalamus,
Nucleus subthalamicus, Hirnrinde, Medulla oblongata, Kleinhirn,
Hinterhauptpol, Stirnlappen, Schläfenlappen, Putamen, Nucleus
caudatus, Corpus callosum, Substantia nigra), Rückenmark, Hypophyse, Magen,
Bauchspeicheldrüse,
Niere, Leber, Geschlechtsdrüse,
Schilddrüse, Gallenblase,
Knochenmark, Nebenniere, Haut, Muskel, Lunge, Magen-Darm-Trakt (beispielsweise
Dickdarm und Dünndarm),
Blutgefäß, Herz,
Thymusdrüse,
Milz, Glandula submandibularis, peripheres Blut, pheripherer Hämozyt, Prostata,
Testikel, Hoden, Eierstock, Plazenta, Uterus, Knochen, Gelenk, Skelettmuskel
usw. (speziell Gehirn und Gehirnregion) usw.; das Peptid oder Protein
kann ebenso ein synthetisches Peptid oder ein synthetisches Protein
sein.
-
Das
Peptid der Erfindung umfaßt
Peptide, die bevorzugt vom Menschen oder anderen Säugern abgeleitet
sind, stärker
bevorzug vom Menschen abgeleitet sind.
-
Beispiele
der Aminosäuresequenz,
die im wesentlichen dieselbe Aminosäuresequenz wie die Aminosäuresequenz
aufweist, die durch SEQ ID NR. 1 dargestellt ist, umfassen eine
Aminosäuresequenz
mit mindestens etwa 90 % Homologie, bevorzugt mindestens etwa 95
% Homologie, und stärker
bevorzugt mindestens etwa 98 % Homologie, zu der Aminosäuresequenz,
die durch SEQ ID NR. 1 dargestellt ist.
-
Bevorzugte
Beispiele der Proteine mit im wesentlichen derselben Aminosäuresequenz
wie die Aminosäuresequenz,
die durch SEQ ID NR. 1 dargestellt ist, sind Proteine mit im wesentlichen
derselben Aminosäuresequenz
wie die Aminosäuresequenz,
die durch SEQ ID NR. 1 dargestellt ist, und mit einer Eigenschaft,
die im wesentlichen äquivalent
zu der der Aminosäuresequenz
ist, die durch SEQ ID NR. 1 dargestellt ist, usw.
-
Bevorzugte
Beispiele des gegenwärtigen
Proteins der Erfindung, das dieselbe oder im wesentlichen dieselbe
Aminosäuresequenz
wie die Aminosäuresequenz
enthält,
die durch SEQ ID NR. 1 dargestellt ist, sind Proteine, die dieselbe
oder im wesentlichen dieselbe Aminosäuresequenz wie die Aminosäuresequenz
enthalten, die durch SEQ ID NR. 1 dargestellt ist, und mit einer
Aktivität,
die im wesentlichen zu der der Aminosäuresequenz äquivalent ist, die durch SEQ
ID NR. 1 dargestellt ist, usw.
-
Die
im wesentlichen äquivalente
Aktivität
bezieht sich auf beispielsweise eine Ligandenbindungsaktivität, eine
Signaltransduktionsaktivität
usw. Der Ausdruck im wesentlichen äquivalent wird in der Bedeutung verwendet,
daß die
Natur dieser Aktivitäten äquivalent
ist. Des halb ist es bevorzugt, daß diese Aktivitäten, wie eine
Ligandenbindungsaktivität,
eine Signaltransduktionsaktivität
usw., in der Wirksamkeit äquivalent
sind (beispielsweise etwa das 0,5-bis etwa 2fache), und es ist zulässig, daß sogar
Unterschiede in den Eigenschaften, wie die Stärke dieser Aktivitäten und
Molekulargewicht des Proteins, vorliegen.
-
Diese
Aktivitäten,
wie eine Ligandenbindungsaktivität,
eine Signaltransduktionsaktivität
usw., können gemäß den öffentlich
bekannten Verfahren analysiert werden, und können ebenso beispielsweise
durch die Assayverfahren oder die Screening-Verfahren, die später beschrieben
werden, analysiert werden.
-
Hierin
nachstehend wird das Protein, das die Aminosäuresequenz enthält, die
durch SEQ ID NR. 1 dargestellt ist, manchmal als ZAQ bezeichnet.
-
Das
gegenwärtige
Protein, das eingesetzt werden kann, umfaßt Proteine, umfassend (i)
eine Aminosäuresequenz,
dargestellt durch SEQ ID NR. 1, von der 1 oder 2 mehr (bevorzugt
ungefähr
1 bis 30, stärker bevorzugt
ungefähr
1 bis 20 und am stärksten
bevorzugt mehrere (1 oder 2)) Aminosäuren gelöscht werden; (ii) eine Aminosäuresequenz,
dargestellt durch SEQ ID NR. 20 oder SEQ ID NR. 21, zu der 1 oder
2 mehr (bevorzugt ungefähr
1 bis 30, stärker
bevorzugt ungefähr
1 bis 20 und am stärksten
bevorzugt mehrere (1 oder 2)) Aminosäuren hinzugefügt werden;
(iii) eine Aminosäuresequenz,
dargestellt durch NR. 20 oder SEQ ID NR. 21, in der 1 oder 2 mehr
(bevorzugt ungefähr
1 bis 30, stärker
bevorzugt ungefähr
1 bis 20 und am stärksten bevorzugt
mehrere (1 oder 2)) Aminosäuren
durch andere Aminosäuren
ersetzt werden; und (iv) eine Kombination der obigen Aminosäuresequenzen,
usw.
-
Beispiele
der Aminosäuresequenz,
die im wesentlichen dieselbe Aminosäuresequenz wie die Aminosäuresequenz
aufeist, die durch SEQ ID NR. 36 dargestellt ist, umfassen eine
Aminosäuresequenz
mit mindestens etwa 90 % Homologie, bevorzugt mindestens etwa 95
% Homologie, und stärker
bevorzugt mindestens etwa 98 % Homologie, zu der Aminosäuresequenz,
die durch SEQ ID NR. 36 dargestellt ist.
-
Bevorzugte
Beispiele von Proteinen, die im wesentlichen dieselbe Aminosäuresequenz
wie die Aminosäuresequenz
enthalten, die durch. SEQ ID NR. 36 dargestellt ist, umfassen Proteine
mit im wesentlichen derselben Aminosäuresequenz wie die Aminosäuresequenz,
die durch SEQ ID NR. 36 dargestellt ist, und mit einer Eigenschaft,
die im wesentlichen zu der der Aminosäuresequenz äquivalent ist, die durch SEQ
ID NR. 36 dargestellt ist, usw.
-
Bevorzugte
Beispiele des Proteins, das dieselbe oder im wesentlichen dieselbe
Aminosäuresequenz wie
die Aminosäuresequenz
enthält,
die durch SEQ ID NR. 36 dargestellt ist, umfassen Proteine, die
dieselbe oder im wesentlichen dieselbe Aminosäuresequenz wie die Aminosäuresequenz
enthalten, die durch SEQ ID NR. 36 dargestellt ist, und eine Aktivität aufweisen,
die im wesentlichen zu der der Aminosäuresequenz äquivalent ist, die durch SEQ
ID NR. 36 dargestellt ist, usw., insbesondere Proteine, die in WO
98/46620 beschrieben sind, usw.
-
Beispiele
der Aminosäuresequenz,
die im wesentlichen dieselbe Aminosäuresequenz wie die Aminosäuresequenz
enthält,
die durch SEQ ID NR. 40 dargestellt ist, umfassen eine Aminosäuresequenz
mit mindestens etwa 97 % Homologie, bevorzugt mindestens etwa 98
% Homologie, stärker
bevorzugt mindestens etwa 99 % Homologie, und am stärksten bevorzugt
mindestens etwa 99,5 % Homologie, zu der Aminosäuresequenz, die durch SEQ ID
NR. 40 dargestellt ist, usw.
-
Bevorzugte
Beispiele der Proteine, die im wesentlichen dieselbe Aminosäuresequenz
wie die Aminosäuresequenz
enthalten, die durch SEQ ID NR. 40 dargestellt ist, sind Proteine,
die im wesentlichen dieselbe Aminosäuresequenz wie die Aminosäuresequenz
enthalten, die durch SEQ ID NR. 40 dargestellt ist, und eine Eigenschaft
aufweisen, die im wesentlichen zu der der Aminosäuresequenz äquivalent ist, die durch SEQ
ID NR. 40 dargestellt ist, usw.
-
Bevorzugte
Beispiele des Proteins, das dieselbe oder im wesentlichen dieselbe
Aminosäuresequenz wie
die Aminosäuresequenz
enthält,
die durch SEQ ID NR. 40 dargestellt ist, umfassen Proteine, die
dieselbe oder im wesentlichen dieselbe Aminosäuresequenz wie die Aminosäuresequenz
enthalten, die durch SEQ ID NR. 40 dargestellt ist, und eine Aktivität aufweisen,
die im wesentlichen zu der der Aminosäuresequenz äquivalent ist, die durch SEQ
ID NR. 40 dargestellt ist, usw.
-
Beispiele
der Aminosäuresequenz,
die im wesentlichen dieselbe Aminosäuresequenz wie die Aminosäuresequenz
aufweist, die durch SEQ ID NR. 47 dargestellt ist, umfassen eine
Aminosäuresequenz
mit mindestens etwa 95 % Homologie, bevorzugt mindestens etwa 96
% Homologie, und stärker
bevorzugt mindestens etwa 97 % Homologie, und am stärksten bevorzugt
mindestens etwa 98 % Homologie, zu der Aminosäuresequenz, die durch SEQ ID
NR. 47 dargestellt ist, usw.
-
Bevorzugte
Beispiele der Proteine, die im wesentlichen dieselben Aminosäuresequenz
wie die Aminosäuresequenz
enthalten, die durch SEQ ID NR. 47 dargestellt ist, sind Proteine,
die im wesentlichen dieselbe Aminosäuresequenz wie die Aminosäuresequenz
enthalten, die durch SEQ ID NR. 47 dargestellt ist, und eine Eigenschaft
aufweisen, die im wesentlichen zu der der Aminosäuresequenz äquivalent ist, die durch SEQ ID
NR. 47 dargestellt ist, usw.
-
Bevorzugte
Beispiele des Proteins, das dieselbe oder im wesentlichen dieselbe
Aminosäuresequenz wie
die Aminosäuresequenz
enthält,
die durch SEQ ID NR. 47 dargestellt ist, sind Proteine, die dieselbe
oder im wesentlichen dieselbe Aminosäuresequenz wie die Aminosäuresequenz
enthalten, die durch SEQ ID NR. 47 dargestellt ist, und eine Aktivität aufweisen,
die im wesentlichen zu der der Aminosäuresequenz äquivalent ist, die durch SEQ
ID NR. 47 dargestellt ist, usw.
-
Beispiele
der Aminosäuresequenz,
die im wesentlichen dieselbe Aminosäuresequenz wie die Aminosäuresequenz
aufweist, die durch SEQ ID NR. 48 dargestellt ist, umfassen eine
Aminosäuresequenz
mit mindestens etwa 95 % Homologie, bevorzugt mindestens etwa 96
% Homologie, und stärker
bevorzugt mindestens etwa 97 % Homologie, und am stärksten bevorzugt
mindestens etwa 98 % Homologie, zu der Aminosäuresequenz, die durch SEQ ID
NR. 48 dargestellt ist, usw.
-
Bevorzugte
Beispiele der Proteine, die im wesentlichen dieselbe Aminosäuresequenz
wie die Aminosäuresequenz
enthalten, die durch SEQ ID NR. 48 dargestellt ist, sind Proteine,
die im wesentlichen dieselbe Aminosäuresequenz wie die Aminosäuresequenz
enthalten, die durch SEQ ID NR. 48 dargestellt ist, und eine Eigenschaft
aufweisen, die im wesentlichen zu der der Aminsäuresequenz äquivalent ist, die durch SEQ
ID NR. 48 dargestellt ist, usw.
-
Bevorzugte
Beispiele des Proteins, das dieselbe oder im wesentlichen dieselbe
Aminosäuresequenz wie
die Aminosäuresequenz
enthält,
die durch SEQ ID NR. 48 dargestellt ist, sind Proteine, die dieselbe
oder im wesentlichen dieselbe Aminosäuresequenz wie die Aminosäu resequenz
enthalten, die durch SEQ ID NR. 48 dargestellt ist, und eine Aktivität aufweisen,
die im wesentlichen zu der der Aminsäuresequenz äquivalent ist, die durch SEQ
ID NR. 48 dargestellt ist, usw.; besonders bevorzugt sind Proteine,
die in Biochem. Biophys. Acta, 1491, 369–375, 2000 beschrieben sind,
usw.
-
Beispiele
der Aminosäuresequenz,
die im wesentlichen dieselbe Aminosäuresequenz wie die Aminosäuresequenz
aufweist, die durch SEQ ID NR. 49 dargestellt ist, umfassen eine
Aminosäuresequenz
mit mindestens etwa 95 % Homologie, bevorzugt mindestens etwa 96
Homologie, und stärker
bevorzugt mindestens etwa 97 % Homologie, und am stärksten bevorzugt
mindestens etwa 98 % Homologie, zu der Aminosäuresequenz, die durch SEQ ID
NR. 49 dargestellt ist.
-
Bevorzugte
Beispiele der Proteine, die im wesentlichen dieselbe Aminosäuresequenz
wie die Aminosäuresequenz
enthalten, die durch SEQ ID NR. 49 dargestellt ist, sind Proteine,
die im wesentlichen dieselbe Aminosäuresequenz wie die Aminosäuresequenz
enthalten, die durch SEQ ID NR. 49 dargestellt ist, und eine Eigenschaft
aufweisen, die im wesentlichen zu der der Aminsäuresequenz äquivalent ist, die durch SEQ
ID NR. 49 dargestellt ist, usw.
-
Bevorzugte
Beispiele des Proteins, das dieselbe oder im wesentlichen dieselbe
Aminosäuresequenz wie
die Aminosäuresequenz
enthält,
die durch SEQ ID NR. 49 dargestellt ist, sind Proteine, die dieselbe
oder im wesentlichen dieselbe Aminosäuresequenz wie die Aminosäuresequenz
enthalten, die durch SEQ ID NR. 49 dargestellt ist, und eine Aktivität aufweisen,
die im wesentlichen zu der der Aminsäuresequenz äquivalent ist, die durch SEQ
ID NR. 49 dargestellt ist, usw.; besonders bevorzugt sind Proteine,
die in WO 98/46620 beschrieben sind, usw.
-
Die
im wesentlichen äquivalente
Aktivität
bezieht sich beispielsweise auf eine Ligandenbindungsaktivität, eine
Signaltransduktionsaktivität
usw. Der Ausdruck im wesentlichen äquivalent wird in der Bedeutung verwendet,
daß die
Natur dieser Aktivitäten äquivalent
ist. Deshalb ist es bevorzugt, daß diese Aktivitäten, wie eine
Ligandenbindungsaktivität,
eine Signaltransduktionsaktivität
usw. in der Wirksamkeit äquivalent
sind (beispielsweise etwa das 0,5-bis etwa 2fache), und es ist zulässig, daß sogar
Unterschiede in den Eigenschaften, wie die Stärke dieser Aktivitäten und
Molekulargewicht des Proteins, vorliegen.
-
Die
Aktivitäten,
wie eine Ligandenbindungsaktivität,
eine Signaltransduktionsaktivität,
usw. kann gemäß den öffentlich
bekannten Verfahren analysiert werden.
-
Beispiele
der Aminosäuresequenz,
die im wesentlichen dieselbe Aminosäuresequenz wie die Aminosäuresequenz
aufweist, die durch SEQ ID NR. 34 dargestellt ist, umfassen eine
Aminosäuresequenz
mit mindestens etwa 60 % Homologie, bevorzugt mindestens etwa 70
% Homologie, stärker
bevorzugt mindestens etwa 80 % Homologie, und am stärksten bevorzugt
mindestens etwa 90 % Homologie, zu der Aminosäuresequenz, die durch SEQ ID
NR. 34 dargestellt ist, usw.
-
Bevorzugte
Beispiele des Peptids, das dieselbe oder im wesentlichen dieselbe
Aminosäuresequenz wie
die Aminosäuresequenz
enthält,
die durch SEQ ID NR. 34 dargestellt ist, sind ein Peptid, das dieselbe
oder im wesentlichen dieselbe Aminosäuresequenz wie die Aminosäuresequenz
enthält,
die durch SEQ ID NR. 34 dargestellt ist, und eine Aktivität aufweist,
die im wesentlichen zu der der Aminosäuresequenz äquivalent ist (beispielsweise
eine kontraktile Aktivität
im Krummdarm, eine kontraktile Aktivität im distalen Dickdarm, eine Relaxationsaktivität im proximalen
Dickdarm usw.), die durch SEQ ID NR. 34 dargestellt ist, usw.; insbesondere
MIT1, das später
beschrieben wird, und so weiter.
-
In
der gesamten vorliegende Beschreibung werden die Peptide und Proteine
gemäß der konventionellen
Weise zum Beschreiben von Peptiden dargestellt, das heißt, der
N-Terminus (Aminoterminus) auf der linken und der C-Terminus (Carboxylterminus)
auf der rechten Seite. In den gegenwärtigen Proteinen, einschließlich den
Proteinen, die die durch SEQ ID NR. 1 gezeigte Aminosäuresequenz
enthalten, kann der C-Terminus eine Carboxylgruppe (-COOH), ein
Carboxylat (-COO–), ein Amid (-CONH2) oder ein Ester (-COOR) sein.
-
Hierin
umfassen Beispiele der Estergruppe, die durch R gezeigt wird, eine
C1-6-Alkylgruppe, wie Methyl, Ethyl, n-Propyl,
Isopropyl, n-Butyl usw.; eine C3-8-Cycloalkylgruppe,
wie Cyclopentyl, Cyclohexyl usw.; eine C6-12-Arylgruppe,
wie Phenyl, α-Naphthyl
usw.; eine C7-14-Aralkylgruppe, wie eine Phenyl-C1-2-alkylgruppe, z.B., Benzyl, Phenethyl
usw.; eine α-Naphthyl-C1-2-alkylgruppe, wie α-Naphthylmethyl usw.; und dergleichen. Außerdem kann Pivaloyloxymethyl
oder dergleichen, das weitgehend als ein Ester zur oralen Verabreichung verwendet
wird, ebenso verwendet werden.
-
Wo
das Peptid der Erfindung Carboxylgruppe (oder ein Carboxylat) an
einer anderen Stelle als den C-Terminus enthält, kann es amidiert oder verestert
werden, und ein solches Amid oder solcher Ester wird ebenso in das
Peptid/Protein der Erfindung einbezogen. Die Estergruppe kann dieselbe
Gruppe sein, wie die, die in bezug auf den obigen C-terminalen Ester
beschrieben wird.
-
Außerdem umfassen
Beispiele des Peptids der Erfindung Varianten des obigen Peptids/Proteins,
wobei die Aminogruppe an dem N-Terminus (z.B. Methioninrest) des
Peptids mit einer Schutzgruppe geschützt wird (z.B. eine C1-6-Acylgruppe, wie eine C2-6-Alkanoylgruppe, z.B.
Formylgruppe, Acetylgruppe usw.); die, wobei die N-terminale Region
in vivo gespalten wird und die so gebildete Glutamylgruppe pyroglutaminiert
wird; die, wobei ein Substituent (z.B. -OH, -SH, Aminogruppe, Imidazolgruppe,
Indolgruppe, Guanidinogruppe usw.) an der Seitenkette einer Aminosäure in dem
Molekül
mit einer geeigneten Schutzgruppe geschützt wird (z.B. eine C1-6-Acylgruppe, wie eine C2-6-Alkanoylgruppe,
z.B. Formylgruppe, Acetylgruppe usw.), oder konjugierte Peptide/konjugierte
Proteine, wie Glycopeptide/Glycoproteine mit Zuckerketten.
-
Spezielle
Beispiele des Peptids der Erfindung umfassen vom Menschen abgeleitete
(stärker
bevorzugt vom menschlichen Gehirn abgeleitete) Peptide, bestehend
aus der Aminosäuresequenz,
die durch SEQ ID NR. 20 dargestellt ist, vom Menschen abgeleitete
(stärker
bevorzugt vom menschlichen Gehirn abgeleitete) Peptide, bestehend
aus der Aminosäuresequenz,
die durch SEQ ID NR. 21 dargestellt ist, usw. Stärker bevorzugt sind es vom
Menschen abgeleitete Peptide, bestehend aus der Aminosäuresequenz,
die durch SEQ ID NR. 21 dargestellt ist.
-
Spezielle
Beispiele des gegenwärtigen
Proteins umfassen vom Menschen abgeleitete (bevorzugt vom menschlichen
Gehirn abgeleitete) Proteine, die die Aminosäuresequenz enthalten, die durch
SEQ ID NR. 1 dargestellt ist, usw.
-
Jedes
Teilpeptid, das für
das gegenwärtige
Protein beschrieben ist, kann als das Teilpeptid des gegenwärtigen Proteins
verwendet werden (hierin nachstehend manchmal nur als gegen wärtiges Teilpeptid
bezeichnet). Beispielsweise kann ein Teil des gegenwärtigen Proteinmoleküls, der
auf der Außenseite
einer Zellmembran oder dergleichen exponiert wird, verwendet werden,
so lange wie er eine im wesentlichen äquivalente Rezeptorbindungsaktivität aufweist.
-
Speziell
ist das Teilpeptid des Proteins, das die Aminosäuresequenz enthält, die
durch SEQ ID NR. 1 dargestellt ist, ein Peptid, das die Teile enthält, die
als extrazelluläre
Domänen
(hydrophile Domänen)
in der hydrophoben Auswertungsanalyse, wie in 7 gezeigt,
analysiert worden sind. Ein Peptid, das einen hydrophoben Domänenteil
enthält,
kann ebenso verwendet werden. Außerdem kann das Peptid, das
jede Domäne separat
enthält,
verwendet werden, aber das Peptid kann mehrere Domänen zusammen
enthalten.
-
Die
Anzahl an Aminosäuren
in dem gegenwärtigen
Teilpeptid beträgt
mindestens 20, bevorzugt mindestens 50 und stärker bevorzugt mindestens 100
Aminosäuren
in der Aminosäuresequenz,
die das gegenwärtige
oben beschriebene Protein bildet, und Peptide mit der Aminosäuresequenz
mit solchen Anzahlen an Aminosäuren
usw. sind bevorzugt.
-
Die
im wesentlichen selbe Aminosäuresequenz
umfaßt
eine Aminosäuresequenz
mit mindestens etwa 50 % Homologie, bevorzugt mindestens etwa 70
% Homologie, stärker
bevorzugt mindestens etwa 80 % Homologie, am stärksten bevorzugt mindestens
etwa 90 % Homologie und am stärksten
bevorzugt mindestens etwa 95 % Homologie, zu diesen Aminosäuresequenzen.
-
Hierin
wird der Ausdruck „im
wesentlichen äquivalente
Ligandenbindungsaktivität" in derselben Bedeutung,
wie oben definiert, verwendet. Die „im wesentlichen äquivalente
Ligandenbindungsaktivität" kam durch eine Modifikation
von öffentlich
bekannten Verfahren analysiert werden.
-
Das
Teilpeptid des Proteins, das dieselbe oder im wesentlichen dieselbe
Aminosäuresequenz
wie die Aminosäuresequenz
enthält,
die durch SEQ ID NR. 36; SEQ ID NR. 40; SEQ ID NR. 47; SEQ ID NR.
48 oder SEQ ID NR. 49 dargestellt ist, kann jedes Teilpeptid sein,
so lange wie es das zuvor genannte Teilpeptid des Proteins ist,
das dieselbe oder im wesentlichen dieselbe Aminosäuresequenz
wie die Aminosäuresequenz
enthält,
die durch SEQ ID NR. 36; SEQ ID NR. 40, SEQ ID NR; 47, SEQ ID NR.
48 oder SEQ ID NR. 49 dargestellt ist, und in den Proteinmolekülen werden
beispielsweise die mit einer Seite, die sich außerhalb der Zellmembran erstreckt
und im wesentlichen äquivalente
Ligandenbindungsaktivität
aufweist, eingesetzt. Die Anzahl an Aminosäuren in dem Teilpeptid beträgt mindestens
20, bevorzugt mindestens 50 und stärker bevorzugt mindestens 100
Aminosäuren
in der Aminosäuresequenz,
die das oben beschriebene Protein bildet, und Peptide mit der Aminosäuresequenz
von solchen Anzahlen von Aminosäuren
usw. sind bevorzugt. Die im wesentlichen selbe Aminosäuresequenz
umfaßt
eine Aminosäuresequenz
mit mindestens etwa 50 % Homologie, bevorzugt mindestens etwa 70
% Homologie, stärker
bevorzugt mindestens etwa 80 % Homologie, am stärksten bevorzugt mindestens
etwa 90 % Homologie und am stärksten
bevorzugt mindestens etwa 95 % Homologie, zu diesen Aminosäuresequenzen.
-
Als
die Salze des Peptids der Erfindung und des gegenwärtigen Proteins
oder seines Teilpeptids usw. sind physiologisch akzeptable Säureadditionssalze
besonders bevorzugt. Beispiele von solchen Salzen sind Salze mit
anorganischen Säuren
(z.B. Salzsäure,
Phosphorsäure,
Bromwasserstoffsäure
und Schwefelsäure), Salze
mit organischen Säuren
(z.B. Essigsäure,
Ameisensäure,
Propionsäure,
Fumarsäure,
Maleinsäure, Bernsteinsäure, Weinsäure, Zitronensäure, Äpfelsäure, Oxalsäure, Benzoesäure, Methansulfonsäure, Benzolsulfonsäure) und
dergleichen.
-
Das
erfindungsgemäße Peptid
oder gegenwärtige
Protein oder Salze davon sowie die Proteine, die die Aminosäuresequenz
enthalten, die durch SEQ ID NR. 36, SEQ ID NR. 40, SEQ ID NR. 47
dargestellt ist, oder Salze davon können durch ein öffentlich
bekanntes Verfahren hergestellt werden, das verwendet wird, um ein
Peptid/Protein aus oben beschriebenen menschlichen oder anderen
Säugerzellen
oder -geweben zu reinigen. Alternativ können sie ebenso durch Kultivieren
einer Transformante hergestellt werden, die eine DNA trägt, die
das erfindungsgemäße Peptid,
das erfindungsgemäße Protein
oder das Protein, das die Aminosäuresequenz
enthält,
die durch SEQ ID NR. 36, SEQ ID NR. 40, SEQ ID NR. 47 dargestellt
ist, kodiert, wie später beschrieben
wird. Sie können
ebenso durch die später
beschriebene Peptid/Protein-Synthese oder durch eine Modifikation
davon hergestellt werden. Außerdem
kann das Protein, das die Aminosäuresequenz
enthält,
die durch SEQ ID NR. 48 dargestellt ist, oder Salze davon durch
eine Modifikation des Verfahrens, das in Biochem. Biophys. Acta,
1491, 369 – 375,
2000 beschrieben ist, hergestellt werden. Das Protein, das die Aminosäure sequenz
enthält,
die durch SEQ ID NR. 49 dargestellt ist, oder Salze davon kann durch
eine Modifikation des Verfahrens, das in WO 98/46620 beschrieben
ist, hergestellt werden.
-
Wenn
die Peptide/Proteine oder Salze davon aus menschlichem oder Säugergewebe
oder -zellen hergestellt werden, werden die menschlichen oder Säugergewebe
oder -zellen homogenisiert und mit einer Säure oder dergleichen extrahiert,
und das Extrakt wird isoliert und durch eine Kombination von chromatographischen
Techniken, wie Umkehrphasenchromatographie, Ionenaustauschchromatographie
und dergleichen, gereinigt.
-
Um
das Peptid der Erfindung, das gegenwärtige Protein, sein Teilpeptid
oder seine Amide, oder Salze davon zu synthetisieren, können kommerziell
erhältliche
Harze, die für
die Peptid/Protein-Synthese genutzt werden, verwendet werden. Beispiele
von solchen Harzen umfassen Chlormethylharz, Hydroxymethylharz, Benzhydrylaminharz,
Aminomethylharz, 4-Benzyloxybenzylalkoholharz, 4-Methylbenzhydrylaminharz, PAM-Harz,
4-Hydroxymethylmethylphenylacetamidomethylharz, Polyacrylamidharz,
4-(2',4'-Dimethoxyphenylhydroxymethyl)phenoxyharz,
4-(2',4'-Dimethoxyphenyl-Fmoc-aminoethyl)phenoxyharz
usw. Unter Verwendung dieser Harze werden Aminosäuren, worin α-Aminogruppen
und funktionelle Gruppen an den Seitenketten entsprechend geschützt werden,
auf dem Harz in der Reihenfolge der Sequenz des Ziel-Peptids/Proteins gemäß den verschiedenen
Kondensationsverfahren, die in der Technik öffentlich bekannt sind, kondensiert. Am
Ende der Reaktion wird das Peptid/Protein aus dem Harz entfernt
und gleichzeitig werden die Schutzgruppen entfernt. Darnn wird die
intramolekulare Disulfid-Bindungsbildungs-Reaktion in einer stark
verdünnten
Lösung
durchgeführt,
um das Ziel-Peptid/Protein oder Amide davon zu erhalten.
-
Zur
Kondensation der geschützten
oben beschriebenen Aminosäuren
kann eine Vielzahl von Aktivierungsreagenzien für die Peptid/Protein-Synthese
verwendet werden, aber Carbodiimide werden besonders bevorzugt eingesetzt.
Beispiele von solchen Carbodiimiden umfassen DCC, N,N'-Diisopropylcarbodiimid, N-Ethyl-N'-(3-dimethylaminopropyl)carbodiimid
usw. Zur Aktivierung durch diese Reagenzien werden die geschützten Aminosäuren in
Kombination mit einem Racemisierungsinhibitor (z.B. HOBt, HOOBt)
direkt zu dem Harz zugegeben, oder die geschützten Aminosäuren werden
zuvor in Form von symmetrischen Säureanhydriden, HOBt-Estern
oder HOOBt-Estern aktiviert, gefolgt von der Zugabe der so aktivierten
geschützten
Aminosäuren
zu dem Harz.
-
Lösungsmittel,
die zur Verwendung geeignet sind, um die geschützten Aminosäuren zu
aktivieren oder mit dem Harz zu kondensieren, können aus den Lösungsmitteln
ausgewählt
werden, die dafür
bekannt sind, daß sie
für die
Peptid/Protein-Kondensationsreaktionen nützlich sind. Beispiele von
solchen Lösungsmitteln sind
Säureamide,
wie N,N-Dimethylformamid, N,N-Dimethylacetamid, N-Methylpyrrolidon
usw.; halogenierte Kohlenwasserstoffe, wie Methylenchlorid, Chloroform
usw.; Alkohole, wie Trifluorethanol usw.; Sulfoxide, wie Dimethylsulfoxid
usw.; Ether, wie Pyridin, Dioxan, Tetrahydrofuran usw.; Nitrile,
wie Acetonitril, Propionitril usw.; Ester, wie Methylacetat, Ethylacetat
usw.; und geeignete Gemische aus diesen Lösungsmitteln. Die Reaktionstemperatur
wird geeigneterweise aus dem Bereich ausgewählt, der dafür bekannt
ist, daß er
auf die Proteinbindungsreaktionen anwendbar ist, und wird normalerweise
in dem Bereich von ungefähr –20 °C bis 50 °C ausgewählt. Die
aktivierten Aminosäurederivate
werden im allgemeinen in einem Überschuß vom 1,5-
bis 4fachen verwendet. Die Kondensation wird unter Verwendung der
Ninhydrinreaktion kontrolliert; wenn die Kondensation unzureichend
ist, kann die Kondensation durch Wiederholten der Kondensationsreaktion
ohne die Entfernung der Schutzgruppen vollendet werden. Wenn die
Kondensation nach dem Wiederholen der Reaktion noch unzureichend
ist, werden nicht-umgesetzte
Aminosäuren
mit Essigsäureanhydrid
oder Acetylimidazol acetyliert.
-
Beispiele
der Schutzgruppen, die verwendet werden, um die Ausgangsaminogruppen
zu schützen, umfassen
Z, Boc, t-Pentyloxycarbonyl, Isobornyloxycarbonyl, 4-Methoxybenzyloxycarbonyl,
Cl-Z, Br-Z, Adamantyloxycarbonyl, Trifluoracetyl, Phthaloyl, Formyl,
2-Nitrophenylsulfenyl, Diphenylphosphinothioyl, Fmoc usw.
-
Eine
Carboxylgruppe kann durch beispielsweise Alkylveresterung (in der
Form von linearen, verzweigten oder cyclischen Alkylestern der Alkyleinheit,
wie Methyl, Ethyl, Propyl, Butyl, t-Butyl, Cyclopentyl, Cyclohexyl,
Cycloheptyl, Cyclooctyl, 2-Adamantyl usw.), Aralkylveresterung (beispielsweise
Veresterung in Form von Benzylester, 4-Nitrobenzylester, 4-Methoxybenzylester,
4-Chlorbenzylester, Benzhydrylester usw.), Phenacylveresterung,
Benzyloxycarbonylhydrazidierung, t-Butoxycarbonylhydrazidierung,
Tritylhydrazidierung oder dergleichen geschützt werden.
-
Die
Hydroxylgruppe von Serin kann beispielsweise durch ihre Veresterung
oder Veretherung geschützt
werden. Beispiele von Gruppen, die geeigneterweise für die Veresterung
verwendet werden, umfassen eine Niederalkanoylgruppe, wie Acetylgruppe,
eine Aroylgruppe, wie Benzoylgruppe, und eine Gruppe, die von Kohlensäure abgeleitet
ist, wie Benzyloxycarbonylgruppe und Ethoxycarbonylgruppe. Beispiele
einer Gruppe, die geeigneterweise für die Veretherung verwendet
wird, umfassen Benzylgruppe, Tetrahydropyranylgruppe, t-Butylgruppe
usw.
-
Beispiele
von Gruppen zum Schutz der phenolischen Hydroxylgruppe von Tyrosin
umfassen Bzl, Cl2-Bzl, 2-Nitrobenzyl, Br-Z,
t-Butyl usw.
-
Beispiele
von Gruppen, die verwendet werden, um die Imidazoleinheit von Histidin
zu schützen,
umfassen Tos, 4-Methoxy-2,3,6-trimethylbenzolsulfonyl, DNP, Benzyloxymethyl,
Bum, Boc, Trt, Fmoc usw.
-
Beispiele
der aktivierten Carboxylgruppen in den Ausgangsaminosäuren umfassen
die entsprechenden Säureanhydride,
Azide, aktivierten Ester (Ester mit Alkoholen (z.B. Pentachlorphenol,
2,4,5-Trichlorphenol, 2,4-Dinitrophenol, Cyanomethylalkohol, p-Nitrophenol,
HONB, N-Hydroxysuccimid; N-Hydroxyphthalimid, HOBt)). Als die aktivierten
Aminosäuren,
worin die Aminogruppen in dem Ausgangsmaterial aktiviert werden, werden
die entsprechenden Phosphoramide eingesetzt.
-
Um
die Schutzgruppen zu beseitigen (abzuspalten), werden katalytische
Reduktion unter Wasserstoffgasstrom in Gegenwart eines Katalysators,
wie Pd-Ruß oder
Pd-Kohlenstoff eine Säurebehandlung
mit wasserfreiem Fluorwasserstoff, Methansulfonsäure, Trifluormethansulfonsäure oder
Trifluoracetat, oder eine Gemischlösung aus diesen Säuren; eine
Behandlung mit einer Base, wie Diisopropylethylamin, Triethylamin,
Piperidin oder Piperazin; und Reduktion mit Natrium in flüssigem Ammoniak
verwendet. Die Beseitigung durch die oben beschriebene Säurebehandlung
wird im allgemeinen bei einer Temperatur von ungefähr –20 °C bis 40 °C durchgeführt. Bei
der Säurebehandlung
ist es effizient, einen Kationenfänger, wie Anisol, Phenol, Thioanisol,
m-Cresol, p-Cresol, Dimethylsulfid, 1,4-Butandithiol oder 1,2-Ethandithiol
zuzugeben. Außerdem
wird die 2,4-Dinitrophenylgruppe, die als die Schutzgruppe für das Imidazol
von Histidin verwendet wird, durch eine Behandlung mit Thi ophenol
entfernt. Die Formylgruppe, die als die Schutzgruppe des Indols
von Tryptophan verwendet wird, wird durch die zuvor genannte Säurebehandlung
in Gegenwart von 1,2-Ethandithiol oder 1,4-Butandithiol sowie durch
eine Behandlung mit einem Alkali, wie einer verdünnten Natriumhydroxidlösung, verdünntem Ammoniak
usw., beseitigt.
-
Der
Schutz von funktionellen Gruppen, die nicht an der Reaktion der
Ausgangsmaterialien beteiligt sein sollten, die Beseitigung der
Schutzgruppen und die Aktivierung der funktionellen Gruppen, die
an der Reaktion beteiligt sind, können entsprechend aus den öffentlich
bekannten Gruppen und öffentlich
bekannten Mitteln ausgewählt
werden.
-
Bei
einem anderen Verfahren zur Erhaltung der Amide des Peptids/Proteins
wird beispielsweise die α-Carboxylgruppe
der Carboxyl-terminalen Aminosäure
zunächst
durch Amidierung geschützt;
die Peptidkette wird dann von der Aminogruppenseite auf eine gewünschte Länge verlängert. Danach
werden ein Peptid/Protein, bei dem nur die Schutzgruppe der N-terminalen α-Aminogruppe
aus der Peptidkette beseitigt worden ist, und ein Peptid/Protein,
bei dem nur die Schutzgruppe aus der C-terminalen Carboxylgruppe
beseitigt worden ist, hergestellt. Die zwei Peptide/zwei Proteine
werden in einem Gemisch aus den oben beschriebenen Lösungsmitteln
kondensiert. Die Einzelheiten der Kondensationsreaktion sind dieselben,
wie oben beschrieben. Nach dem das geschützte Peptid/geschützte Protein,
das durch Kondensation erhalten wurde, gereinigt wurde, wurden alle
Schutzgruppen durch das oben beschriebene Verfahren beseitigt, um
das gewünschte
rohe Peptid/rohe Protein zu erhalten. Das rohe Peptid/rohe Protein
wird durch verschiedene Reinigungsmittel gereinigt. Lyophilisierung
der Hauptfraktion ergibt das Amid des gewünschten Peptids/Proteins.
-
Um
das veresterte Peptid/Protein herzustellen, wird beispielsweise
die α-Carboxylgruppe
der Carboxyl-terminalen Aminosäure
mit dem gewünschten
Alkohol kondensiert, um den Aminosäureester herzustellen, worauf
die Verfahrensweise folgt, die der Herstellung des amidierten Proteins
oben ähnelt,
wodurch das gewünschte
veresterte Peptid/Protein erhalten wurde.
-
Das
Peptid der Erfindung kann gemäß der öffentlich
bekannten Peptidsynthese hergestellt werden.
-
Das
Teilpeptid des gegenwärtigen
Proteins der Erfindung oder Salze davon kann durch öffentlich
bekannte Verfahren zur Peptidsynthese oder durch Spalten des gegenwärtigen Proteins
mit einer geeigneten Peptidase hergestellt werden.
-
Für die Verfahren
zur Peptidsynthese kann beispielsweise entweder Festphasensynthese
oder Flüssigphasensynthese
verwendet werden. Das heißt,
das Teilpeptid oder die Aminosäuren,
die das Peptid der Erfindung oder das Protein der Erfindung bilden,
werden mit dem verbliebenen Teil kondensiert. Wo das Produkt Schutzgruppen
umfaßt,
werden diese Schutzgruppen entfernt, wodurch das gewünschte Peptid
erhalten wurde. Öffentlich
bekannte Verfahren zur Kondensation und Beseitigung der Schutzgruppen
werden in (1) bis (5) nachstehend beschrieben.
- (1) M. Bodanszky & M. A. Ondetti:
Peptide Synthesis, Interscience Publishers, New York (1966)
- (2) Schroeder & Luebke:
The Peptide, Academic Press, New York (1965)
- (3) Nobuo Izumiya, et al.: Peptide Gosei-no-Kiso to Jikken (Basics
and experiments of peptide synthesis), veröffentlich von Maruzen Co. (1975)
- (4) Haruaki Yajima & Shunpei
Sakakibara: Seikagaku Jikken Koza (Biochemical Experiment) 1, Tanpakushitsu no
Kagaku (Chemistry of Proteins) IV, 205 (1977)
- (5) Haruaki Yajima Hrsg.: Zoku Iyakuhin no Kaihatsu (A sequel
to Development of Pharmaceuticals), Bd. 14, Peptide Synthesis, veröffentlicht
von Hirokawa Shoten
-
Nach
der Beendigung der Reaktion kann das Produkt durch eine Kombination
von konventionellen Reinigungsverfahren, wie Lösungsmittelextraktion, Destillation,
Säulenchromatographie,
Flüssigchromatographie
und Umkristallisierung gereinigt und isoliert werden, wodurch das
Peptid oder das Teilpeptid der Erfindung erhalten wird. Wenn das
Peptid oder das Teilpeptid, das durch die obigen Verfahren erhalten
wurde, in einer freien Form vorliegt, kann das Peptid in ein entsprechendes
Salz durch ein öffentlich
bekanntes Verfahren umgewandelt werden; wenn das Protein in einer
Salzform erhalten wird, kann es in eine freie Form durch ein öffentlich
bekanntes Verfahren umgewandelt werden.
-
Die
DNA, die das Peptid/Protein der Erfindung kodiert, kann jede DNA
sein, so lange wie siei die Basensequenz umfaßt, die das oben beschriebene
Peptid/Protein der Erfindung ko diert. Eine solche DNA kann ebenso
irgendeine der genomischen DNA, genomischen DNA-Bibliothek, cDNA, abgeleitet von den
oben beschriebenen Zellen oder Geweben, cDNA-Bibliothek, abgeleitet von den oben
beschriebenen Zellen oder Geweben, und synthetische DNA sein. Der
Vektor, der für
die Bibliothek verwendet werden soll, kann eine Bakteriophage, Plasmid,
Cosmid, Phagemid und dergleichen sein. Außerdem kann die DNA durch Umkehrtrauskriptase-Polymerase-Kettenreaktion
(hierin nachstehend RT-PCR abgekürzt)
mit gesamter RNA- oder mRNA-Fraktion, hergestellt aus den oben beschriebenen
Zellen oder Geweben, amplifiziert werden.
-
Speziell
kann die DNA, die das Peptid der Erfindung kodiert, irgendeine von
beispielsweise der DNA sein, die die Basensequenz enthält, die
durch SEQ ID NR. 26 oder SEQ ID NR. 27 dargestellt ist.
-
Beispiele
der DNA, die zu der DNA, die die Basensequenz enthält, die
durch SEQ ID NR. 26 dargestellt ist, unter hochstringenten Bedingungen
hybridisierbar ist, umfassen DNAs mit mindestens etwa 60 % Homologie,
bevorzugt mindestens etwa 70 % Homologie und stärker bevorzugt mindestens etwa
80 % Homologie, zu der DNA, die die Basensequenz enthält, die
durch SEQ ID NR. 26 dargestellt ist, usw.
-
Beispiele
der DNA, die zu der DNA, die die Basensequenz enthält, die
durch SEQ ID NR. 27 dargestellt ist, unter hochstringenten Bedingungen
hybridisierbar ist, umfassen DNAs mit mindestens etwa 60 % Homologie,
bevorzugt mindestens etwa 70 % Homologie und stärker bevorzugt mindestens etwa
80 % Homologie, zu der DNA, die die Basensequenz enthält, die
durch SEQ ID NR. 27 dargestellt ist, usw.
-
Ebenso
kann die DNA, die das gegenwärtige
Protein der Erfindung kodiert, irgendeine von beispielsweise einer
DNA, die eine Basensequenz umfaßt,
die durch SEQ ID NR. 2 oder SEQ ID NR. 3 dargestellt ist, und einer
DNA sein, die DNA enthält,
die zu einer DNA, die die Basensequenz enthält, die durch SEQ ID NR. 2
oder SEQ ID NR. 3 dargestellt ist, unter hochstringenten Bedingungen
hybridisierbar ist, und ein Protein kodiert, das eine Aktivität aufweist
(z.B. eine Ligandenbindungsaktivität, eine Signaltransduktionsaktivität usw.), die
im wesentlichen zu der des Proteins der Erfindung äquivalent
ist.
-
Beispiele
der DNA, die zu einer DNA, die die Basensequenz enthält, die
durch SEQ ID NR. 2 oder SEQ ID NR. 3 dargestellt ist, unter hochstringenten
Bedingungen hybridisierbar ist, umfassen DNAs mit mindestens etwa
90 % Homologie, bevorzugt mindestens etwa 95 % Homologie und stärker bevorzugt
mindestens etwa 98 % Homologie, zu der Basensequenz, die durch SEQ
ID NR. 2 oder SEQ ID NR. 3 dargestellt ist, usw.
-
Die
Hybridisierung kann durch öffentlich
bekannte Verfahren oder durch Modifikationen davon beispielsweise
gemäß dem Verfahren,
das in Molecular Cloning, 2nd. (J. Sambrook et al., Cold Spring
Harbor Lab. Press, 1989) beschrieben ist, durchgeführt werden
usw. Eine kommerziell erhältliche
Bibliothek kann ebenso gemäß den Instruktionen
des beiliegenden Herstellerprotokolls verwendet werden. Die Hybridisierung kann
bevorzugt unter hochstringenten Bedingungen durchgeführt werden.
-
Die
hochstringenten Bedingungen, die hierin verwendet werden, sind beispielsweise
eine Natriumkonzentration von ungefähr 19 bis 40 mM, bevorzugt
ungefähr
19 bis 20 mM bei einer Temperatur von ungefähr 50 bis 70 °C, bevorzugt
ungefähr
60 bis 65 °C.
Insbesondere sind Hybridisierungsbedingungen bei einer Natriumkonzentration
von etwa 19 mM bei einer Temperatur von etwa 65 °C am stärksten bevorzugt.
-
Spezieller
gibt es für
die DNA, die das Peptid kodiert, das die Aminosäuresequenz enthält, die
durch SEQ ID NR. 20 dargestellt ist, eine DNA, die die Basensequenz
enthält,
die durch SEQ ID NR. 26 dargestellt ist; gibt es für die DNA,
die das Peptid kodiert, das die Aminosäuresequenz enthält, das
durch SEQ ID NR. 21 dargestellt ist, eine DNA, die die Basensequenz
enthält,
die durch SEQ ID NR. 27 dargestellt ist; gibt es für die DNA,
die das Peptid kodiert, die die Aminosäuresequenz enthält, die
durch SEQ ID NR. 22 dargestellt ist, eine DNA, die die Basensequenz
enthält,
die durch SEQ ID NR. 28 dargestellt ist; und gibt es für die DNA,
die das Peptid kodiert, das die Aminosäuresequenz enthält, die
durch SEQ ID NR. 23 dargestellt ist, eine DNA, die die Basensequenz
enthält,
die durch SEQ ID NR. 29 dargestellt ist.
-
Für die DNA,
die das Protein kodiert, die die Aminosäuresequenz enthält, die
durch SEQ ID NR. 1 dargestellt ist, gibt es eine DNA, die die Basensequenz
enthält,
die durch SEQ ID NR. 2 oder SEQ ID NR. 3 dargestellt ist.
-
Die
Nukleotide (Oligonukleotid), die die Basensequenz oder einen Teil
der Basensequenz umfassen, die komplementär zu der DNA ist, die das Peptid
der Erfindung kodiert, werden in der Bedeutung verwendet, daß nicht
nur die DNA, die das Peptid der Erfindung kodiert, sondern auch
RNA einbezogen ist.
-
Antisense(oligo)nukleotide
(Nukleinsäure),
die die Replikation oder Expression des Gens für das Peptid der Erfindung
inhibieren können,
können
konstruiert und synthetisiert sein, basierend auf der geklonten oder
bestimmten Basensequenzinformation der DNA, die das Peptid kodiert.
Ein solches (Oligo)nukleotid (Nukleinsäure) ist zur RNA des Gens für das Peptid
der Erfindung hybridisierbar, wodurch die Synthese oder Funktion
der RNA inhibiert wird, oder kann die Genexpression des Peptids
der Erfindung über
Interaktion mit RNA, die mit dem Peptid der Erfindung verbunden
ist, modulieren oder kontrollieren. (Oligo)nukleotide, die zu den ausgewählten Sequenzen
von RNA, die mit dem Peptid der Erfindung verbunden ist, komplementär sind,
und (Oligo)nukleotide, die speziell mit der RNA, die mit dem Peptid
der Erfindung verbunden sind, hybridisierbar sind, sind beim Modulieren
oder Kontrollieren der Genexpression des Peptids der Erfindung in
vivo und in vitro nützlich
und zur Behandlung oder Diagnose von Krankheiten usw. nützlich.
-
Der
Ausdruck „entsprechend" wird in der Bedeutung
verwendet, die homolog zu oder komplementär zu einer speziellen Sequenz
des Nukleotids oder Basensequenz oder Nukleinsäure, einschließlich Gen,
ist. Der Ausdruck „entsprechend" zwischen Nukleotiden,
Basensequenzen oder Nukleinsäuren
und Peptiden bezieht sich normalerweise auf Aminosäuren eines
Peptids unter Instruktionen, die aus der Sequenz von Nukleotiden
(Nukleinsäuren)
oder ihren Komplements abgeleitet sind. Die 5'-Enden-Haarnadelschleife, die 5'-Enden-6-Basenpaar-Wiederholungen, die
5'-Enden-untranslatierte
Region, das Polypeptid-Translationsinitiationscodon, die Protein-kodierende
Region, das ORF-Translationsinitiationscodon, die 3'-untranslatierte
Region, die 3'-Enden-Palindromregion
und die 3'-Enden-Haarnadelschleife
können
als bevorzugte Targetregionen ausgewählt werden, obwohl jede andere
Region ebenso als ein Target in Genen des Peptids der Erfindung ausgewählt werden
kann.
-
Die
Beziehung zwischen den targetierten Nukleinsäuren und den (Oligo)nukleotiden,
die komplementär
zu mindestens einem Teil des Targets sind, speziell die Beziehung
zwischen dem Target und den (Oligo)nukleotiden, die zu dem Target
hybridisierbar sind, kann als „An tisense" bezeichnet werden.
Beispiele von Antisense(oligo)nukleotiden umfassen Polydeoxynukleotide,
die 2-Deoxy-D-ribose tragen, Polydeoxynukleotide, die D-Ribose tragen,
jeder andere Typ von Polynukleotiden, die N-Glycoside einer Purin-
oder Pyrimidinbase sind, oder andere Polymere, die Nicht-Nukleotidhautpketten
umfassen (z.B. Proteinnukleinsäuren
und kommerziell erhältliche
synthetische sequenzspezifische Nukleinsäurepolymere) oder andere Polymere,
die Nichtstandardverknüpfungen
umfassen (vorausgesetzt, daß die
Polymere Nukleotide mit einer solchen Konfiguration enthalten, die
die Basenpaarung oder Basenstapelung erlauben, wie sie in DNA oder
RNA gefunden werden können)
usw. Die Antisense-Polynukleotide
können
doppelsträngige
DNA, einsträngige
DNA, doppelsträngige
RNA, einsträngige
RNA oder ein DNA : RNA-Hybrid sein, und können außerdem nicht-modifizierte Polynukleotide
oder nicht-modifizierte Oligonukleotide umfassen, die mit öffentlich
bekannten Modifikationstypen, beispielsweise die mit Markierungen,
die in der Technik bekannt sind, die mit caps-methylierten Polynukleotiden,
die mit Substitution von einem oder mehreren natürlich vorkommenden Nukleotiden
durch ihre Analoga, die mit intramolekularen Modifikationen von
Nukleotiden, wie die mit ungeladenen Verknüpfungen (z.B. Methylphosphonate,
Phosphotriester, Phosphoramidate, Carbamate usw.) und die mit geladenen
Verknüpfungen
oder Schwefel-umfassenden Verknüpfungen
(z.B. Phosphorthioate, Phosphordithioate usw.), die mit Seitenkettengruppen,
wie Proteine (Nukleasen, Nukleaseinhibitoren, Toxine, Antikörper, Signalpeptide,
Poly-L-lysin usw.), Saccharide (z.B. Monosaccharide usw.), die mit
Intercalatoren (z.B. Acridin, Psoralen usw.), die, umfassend Chelatoren
(z.B. Metalle, radioaktive Metalle, Bor, oxidative Metalle usw.),
die, umfassend Alkylierungsmittel, die mit modifizierten Verknüpfungen
(z.B. α-anomere
Nukleinsäuren
usw.), und dergleichen. Hierin sind unter den Ausdrücken „Nukleosid", „Nukleotid" und „Nukleinsäure" Einheiten zu verstehen,
die nicht nur die Purin- und Pyrimidinbasen enthalten, sondern ebenso
andere heterocyclische Basen, die modifiziert worden sind. Diese
Modifikationen können
methylierte Purine und Pyrimidine, acylierte Purine und Pyrimidine
und andere heterocyclische Ringe umfassen. Modifizierte Nukleotide
und modifizierte Nukleotide umfassen ebenso Modifikationen auf der
Zuckereinheit, wobei beispielsweise eine oder mehrere Hydroxylgruppen
gegebenenfalls mit einem Halogenatom(en), einer aliphatischen Gruppe(n)
usw. substituiert sein kann, oder zu den entsprechenden funktionellen
Gruppen, wie Ethern, Aminen oder dergleichen, umgewandelt werden kann/können.
-
Die
Antisense-Nukleinsäure
ist RNA, DNA oder eine modifizierte Nukleinsäure. Spezielle Beispiele der modifizierten
Nukleinsäure
sind Schwefel- und Thiophosphatderivate von Nukleinsäuren und
die, die gegen den Abbau von Polynukleosidamiden oder Oligonukleosidamiden
resistent sind, sind aber nicht darauf beschränkt. Die Antisense-Nukleinsäuren der
vorliegenden Erfindung können
bevorzugt modifiziert werden, basierend auf der folgenden Gestaltung,
das heißt,
durch Erhöhen
der intrazellulären
Stabilität
der Antisense-Nukleinsäure,
Erhöhen
der zellulären
Durchlässigkeit
der Antisense-Nukleinsäure,
Erhöhen
der Affinität
der Nukleinsäure
für den
targetierten codierenden Strang auf ein höheres Niveau, oder Minimieren
der Toxizität,
wenn vorhanden, von der Antisense-Nukleinsäure.
-
Viele
dieser Modifikationen sind in der Technik bekannt, wie in J. Kawakami,
et al., Pharm. Tech. Japan, Bd. 8, S. 247, 1992; Bd. 8, S. 395,
1992; S. T. Crooke, et al. Hrsg., Antisense Research and Applications, CRC
Press, 1993; usw. offenbart.
-
Die
Antisense-Nukleinsäure
kann veränderte
oder modifizierte Zucker, Basen oder Verknüpfungen umfassen. Die Antisense-Nukleinsäure kann
ebenso in einer spezialisierten Form bereitgestellt werden, wie Liposomen,
Mikrokügelchen,
oder kann auf die Gentherapie angewandt werden, oder kann in Kombination
mit angelagerten Einheiten bereitgestellt werden. Diese angelagerten
Einheiten umfassen Polykationen, wie Polylysin, die als Ladungsneutralisatoren
der Phosphathauptkette fungieren, oder hydrophobe Einheiten, wie
Lipide (z.B. Phospholipide, Cholesterole usw.), die die Interaktion
mit Zellmembranen verbessern oder die Aufnahme von Nukleinsäure erhöhen. Bevorzugte
Beispiele der Lipide, die angelagert werden sollen, sind Cholesterole
oder Derivate davon (z.B. Cholesterylchlorameisensäureester,
Cholsäure
usw.). Diese Einheiten können
mit der Nukleinsäure
an den 3'- oder
5'-Enden davon zugeführt werden
und können
ebenso daran durch eine Base, einen Zucker oder intramolekulare
Nukleosidverknüpfung
angelagert sein. Andere Einheiten können Capping-Gruppen sein, die
speziell an den 3'-
oder 5'-Enden der
Nukleinsäure
plaziert sind, um den Abbau durch Nukleasen, wie Exonuklease, RNase,
usw. zu verhindern. Diese Capping-Gruppen umfassen Hydroxylschutzgruppen,
die in der Technik bekannt sind, einschließlich Glykolen wie Polyethylenglykol,
Tetraethylenglykol und dergleichen, sind aber nicht darauf beschränkt.
-
Die
inhibierende Wirkung der Antisense-Nukleinsäure kann unter Verwendung der
Transformante der vorliegenden Erfindung, des Genexpressionssystems
der vorliegenden Erfindung in vivo und in vitro oder des Translationssystems
des Proteins in vivo und in vitro untersucht werden. Die Nukleinsäure kann
auf die Zellen durch eine Vielzahl von öffentlich bekannten Verfahren
angewendet werden.
-
Die
DNA, die das gegenwärtige
Teilpeptid kodiert, kann jede DNA sein, so lange wie sie die Basensequenz
enthält,
die das oben beschriebene gegenwärtige
Teilpeptid kodiert. Die DNA kann ebenso irgendeine von genomischer
DNA, genomischer DNA-Bibliothek, cDNA, abgeleitet aus den oben beschriebenen
Zellen und Geweben, cDNA-Bibliothek, abgeleitet aus den oben beschriebenen
Zellen und Geweben, und synthetischer DNA sein. Der Vektor, der
für die
Bibliothek verwendet werden soll, kann ein Bakteriophage, Plasmid, Cosmid
und Phagemid sein. Die DNA kann ebenso direkt durch Umkehrtranskriptase-Polymerase-Kettenreaktion (hierin
nachstehend als RT-PCR abgekürzt)
unter Verwendung der mRNA-Fraktion,
hergestellt aus den oben beschriebenen Zellen und Geweben, amplifiziert
werden.
-
Speziell
kann die DNA, die das gegenwärtige
Teilpeptid kodiert, irgendeine von beispielsweise einer DNA, die
eine Teilbasensequenz der DNA mit der Basensequenz, die durch SEQ
ID NR. 2 oder SEQ ID NR. 3 dargestellt ist, enthält, oder (2) einer DNA sein,
die eine Teilbasensequenz der DNA mit einer Basensequenz enthält, die
zu der Basensequenz, die durch SEQ ID NR. 2 oder SEQ ID NR. 3 dargestellt
ist, unter hochstringenten Bedingungen hybridisierbar ist, und ein
Protein kodiert, das eine Aktivität aufweist (z.B. eine Ligandenbindungsaktivität, eine
Signaltransduktionsaktivität
usw.), die im wesentlichen zu der des gegenwärtigen Proteins äquivalent
ist.
-
Beispiele
der DNA, die zu einer DNA, die die Basensequenz enthält, die
durch SEQ ID NR. 2 oder SEQ ID NR. 3 dargestellt ist, unter hochstringenten
Bedingungen hybridisierbar ist, umfassen DNAs, die eine Basensequenz
mit mindestens etwa 90 % Homologie, bevorzugt mindestens etwa 95
% Homologie und stärker bevorzugt
mindestens etwa 98 % Homologie, zu der Basensequenz, die durch SEQ
ID NR. 2 oder SEQ ID NR. 3 dargestellt ist, enthalten, usw.
-
Zum
Klonen der DNA, die das Peptid der Erfindung vollständig kodiert,
kann die DNA durch PCR unter Verwendung von synthetischen DNA-Primern,
die einen Teil der Basensequenz einer DNA enthalten, die das Peptid
der Erfindung kodiert, amplifiziert werden, oder die DNA, die in
einen geeigneten Vektor eingeführt
wird, kann durch Hybridisierung mit einem markierten DNA-Fragment
oder synthetischer DNA, die einen Teil oder die vollständige Region
des Peptids der Erfindung kodierieren, ausgewählt werden. Die Hybridisierung
kann beispielsweise gemäß dem Verfahren
durchgeführt
werden, daß in
Molecular Cloning, 2nd, (J. Sambrook et al., Cold Spring Harbor
Lab. Press, 1989) beschrieben ist. Die Hybridisierung kann ebenso
unter Verwendung einer kommerziell erhältlichen Bibliothek gemäß dem Protokoll,
das in den beiliegenden Instruktionen beschrieben ist, durchgeführt werden.
-
Das
Klonen der DNA, die das gegenwärtige
Protein oder sein. Teilpeptid vollständig kodiert (hierin nachstehend
nur als das gegenwärtige
Protein bezeichnet), kann in einer Weise durchgeführt werden,
die dem Klonen der DNA, die das Peptid der Erfindung vollständig kodiert, ähnlich ist.
-
Die
Umwandlung der Basensequenz der DNA kann durch öffentlich bekannte Verfahren,
wie ODA-LA PCR, das Gapped Duplex-Verfahren oder das Kunkel-Verfahren
usw., oder Modifikationen davon, durch PCR oder unter Verwendung
von öffentlich
bekannten Kits, erhältlich
als MutanTM-supper Express Km (hergestellt von
Takara Shuzo Co., Ltd.), MutanTM-K (hergestellt
von Takara Shuzo Co., Ltd.) oder dergleichen bewirkt werden.
-
Die
geklonte DNA, die das Peptid/Protein kodiert, kann als solches in
Abhängigkeit
des Zwecks oder, wenn gewünscht,
nach der Digestion mit einem Restriktionsenzym oder nach der Addition
eines Linkers dazu verwendet werden. Die DNA kann ATG als ein Translationsinitiationscodon
an dem 5'-Ende davon
tragen und kann außerdem
TAA, TGA oder TAG als ein Translationsterminationscodon an dem 3'-Ende davon tragen.
Diese Translationsinitiations- und -terminationscodone können ebenso
unter Verwendung eines geeigneten synthetischen DNA-Adapters zugefügt werden.
-
Der
Expressionsvektor für
das Peptid/Protein kann hergestellt werden, beispielsweise durch
(a) Exzidieren des gewünschten
DNA-Fragments aus der DNA, die das Peptid/Protein der Erfindung
kodiert, und dann (b) Ligieren des DNA-Fragments mit einem geeigneten
Expressionsvektor stromabwärts
eines Promotors in dem Vektor.
-
Beispiele
des Vektors umfassen Plasmide, abgeleitet von E. Coli (z.B. pBR322,
pBR325, pUC12, pUC13), Plasmide, abgeleitet von Bacillus subtilis
(z.B. pUB110, pTP5, pC194), Plasmide, abgeleitet von Hefe (z.B.
pSH19, pSH15), Bakteriophagen, wie λPhage usw., Tierviren, wie Retrovirus,
Vacciniavirus, Baculovirus usw. sowie pA1-11, pXT1, pRc/CMV, pRc/RSV,
pcDNAI/Neo, pcDNA3.1, pRc/CMV2, pRc/RSV (Invitrogen), usw.
-
Der
Promotor, der in der vorliegenden Erfindung verwendet wird, kann
jeder Promotor sein, wenn er mit einem Wirt, der für die Genexpression
verwendet werden soll, gut zusammenpaßt. Bei der Verwendung von
Tierzellen als Wirt umfassen die Beispiele des Promotors SRα-Promotor,
SV40-Promotor, HIV-LTR-Promotor, CMV-Promotor, HSV-TK-Promotor usw.
-
Unter
diesen wird der CMV-Promotor oder SRα-Promotor bevorzugt verwendet.
Wenn der Wirt Bakterien der Gattung Escherichia ist, umfassen bevorzugte
Beispiele des Promotors trp-Promotor, lac-Promotor, recA-Promotor, λPL-Promotor, lpp-Promotor usw. Bei der Verwendung
von Bakterien der Gattung Bacillus als Wirt sind bevorzugte Beispiele
des Promotors SPO1-Promotor, SPO2-Promotor und penP-Promotor. Wenn Hefe
als Wirt verwendet wird, sind bevorzugte Beispiele des Promotors
PHO5-Promotor, PGK-Promotor, GAP-Promotor
und ADH-Promotor. Wenn Insektenzellen als Wirt verwendet werden,
umfassen bevorzugte Beispiele des Promotors Polyhedrin-Promoter
und P10-Promotor.
-
Zusätzlich zu
den vorhergehenden Beispielen kann der Expressionsvektor außerdem gegebenenfalls einen
Enhancer, ein Splicing-Signal, ein Poly-A-Additionssignal, einen
Selektionsmarker, SV40-Replikationsstartpunkt (hierin nachstehend
manchmal als SV40ori abgekürzt)
usw. umfassen. Beispiele des Selektionsmarkers umfassen Dihydrofolatreduktase-
(hierin nachstehend manchmal als dhfr abgekürzt) -Gen [Methotrexat-Resistenz
(MTX-Resistenz)], Ampicillin-Resistenzgen (hierin manchmal als Ampr abgekürzt),
Neomycin-Resistenzgen (hierin nachstehend manchmal als Neor abgekürzt,
G418-Resistenz) usw. Insbesondere kann, wenn dhfr-Gen als der Selektionsmarker
in CHO-(dhfr)-Zellen verwendet wird, die Selektion ebenso auf Thymidin-freien
Medien durchgeführt
werden.
-
Wenn
notwendig, wird eine Signalsequenz, die mit einem Wirt zusammenpaßt, zu dem
N-Terminus des Proteins der Erfindung zugefügt. Beispiele der Signalsequenz,
die verwendet werden kann, sind Pho-A-Signalsequenz, OmpA-Signalsequenz
usw. bei der Verwendung von Bakterien der Gattung Escherichia als
Wirt; α-Amylase-Signalsequenz,
Subtilisin-Signalsequenz,
usw. bei der Verwendung von Bakterien der Gattung Bacillus als Wirt;
MFa-Signalsequenz,
SUC2-Signalsequenz usw. bei der Verwendung von Hefe als Wirt; bzw.
Insulin-Signalsequenz, α-Interferon-Signalsequenz,
Antikörpermolekül-Signalsequenz
usw. bei der Verwendung von Tierzellen als Wirt.
-
Unter
Verwendung des Vektors, der die DNA umfaßt, die das so konstruierte
Peptid/Protein der Erfindung kodiert, können Transformanten hergestellt
werden.
-
Beispiele
des Wirts, der eingesetzt werden kann, sind Bakterien, die zu der
Gattung Escherichia gehören,
Bakterien, die zu der Gattung Bacillus gehören, Hefeinsektenzellen, Insekten
und Tierzellen usw.
-
Spezielle
Beispiele der Bakterien, die zu der Gattung Escherichia gehören, umfassen
Escherichia coli K12 DH1 [Prpc. Natl. Acad. Sci. U.S.A., 60, 160
(1968)], JM103 [Nucleic Acids Research, 9, 309 (1981)], JA221 [Journal
of Molecular Biology, 120, 517 (1978)], HB101 [Journal of Molecular
Biology, 41, 459 (1969)), C600 (Genetics, 39, 440 (1954)], usw.
-
Beispiele
der Bakterien, die zu der Gattung Bacillus gehören, umfassen Bacillus subtilis
MI114 [Gene, 24, 255 (1983)), 207–21 (Journal of Biochemistry,
95, 87 (1984)], usw.
-
Beispiele
von Hefe umfassen Saccharomyces cerevisiae AH22, AH22R–,
NA87-11A, DKD-SD, 20B-12,
Schizosaccharomyces pombe NCYC1913, NCYC2036, Pichia pastoris, usw.
-
Beispiele
von Insektenzellen umfassen für
den Virus AcNPV Spodoptera-frugiperda-Zellen (Sf-Zellen), MG1-Zellen,
abgeleitet vom Mitteldarm von Trichoplusia ni, High FiveTM-Zellen,
abgeleitet vom Ei von Trichoplusia ni, Zellen, abgeleitet von Mamestra
brassicae, Zellen, abgeleitet von Estigmena acrea, usw.; und für den Virus
BmNPV werden Bombyx-mori-N-Zellen
(BmN-Zellen), usw. verwendet. Beispiele der Sf-Zelle, die verwendet
werden kann, sind Sf9-Zellen (ATCC CRL1711) und Sf21-Zellen (beide
Zellen werden in Vaughn, J. L. et al., In Vivo, 13, 213–2l7 (1977),
usw. beschrieben).
-
Als
das Insekt kann beispielsweise eine Larve von Bombyx mori usw. verwendet
werden [Maeda, et al., Nature, 315, 592 (1985)].
-
Beispiele
von Tierzellen umfassen Affenzellen COS-7, Vero, Zellen vom Chinesischen
Hamster CHO (hierin nachstehend als CHO-Zellen bezeichnet), dhfr-Gen-defiziente
Zellen vom Chinesischen Hamster CHO (hierin nachstehend einfach
als CHO(dhfr–)-Zelle
bezeichnet), Maus-L-Zellen, Maus-AtT-20, Maus-Myelomzellen, Ratten-GH3,
Menschen-FL-Zellen, usw.
-
Bakterien,
die zu der Gattung Escherichia gehören, können beispielsweise durch das
Verfahren transformiert werden, das in Proc. Natl. Acad. Sci. U.S.A.,
69, 2110 (1972) oder Gen, 17, 107 (1982), usw. beschrieben ist.
-
Bakterien,
die zu der Gattung Bacillus gehören,
können
beispielsweise durch das Verfahren transformiert werden, das in
Molecular & General
Genetics, 168, 111 (1979), usw. beschrieben ist.
-
Hefe
kann beispielsweise durch das Verfahren transformiert werden, das
in Enzymology, 194, 182–187
(1991), Proc. Natl. Acad. Sci. U.S.A., 75, 1929 (1978), usw. beschrieben
ist.
-
Insektenzellen
oder Insekten können
beispielsweise gemäß dem Verfahren
transformiert werden, das in Bio/Technology, 6, 47–55 (1988),
usw. beschrieben ist.
-
Tierzellen
können
beispielsweise gemäß dem Verfahren
transformiert werden, das in Saibo Kogaku (Cell Engineering), Extraausgabe
8, Shin Saibo Kogaku Jikken Protocol (New Cell Engineering Experimental Protocol),
263–267
(1995), (veröffentlicht
von Shujunsha), oder Virology, 52, 456 (1973) beschrieben ist.
-
Daher
kann die Transformante, transformiert mit dem Expressionsvektor,
der die DNA umfaßt,
die das G-Protein-gekoppelte Rezeptorprotein kodiert, erhalten werden.
-
Wenn
der Wirt Bakterien sind, die zu der Gattung Escherichia oder der
Gattung Bacillus gehören,
kann die Transformante geeigneterweise in einem flüssigen Medium
inkubiert werden, welches Materialien umfaßt, die für das Wachstum der Transformante
erforderlich sind, wie Kohlenstoffquellen, Stickstoffquellen, anorganische
Materialien und so weiter. Beispiele der Kohlenstoffquellen umfassen
Glukose, Dextrin, lösliche
Stärke, Saccharose
usw. Beispiele der Stickstoffquellen umfassen anorganische oder
organische Materialien, wie Ammoniumsalze, Nitratsalze, Maisquellwasser,
Pepton, Casein, Fleischextrakt, Sojabohnenkuchen, Kartoffelextrakt
usw. Beispiele der anorganischen Materialien sind Calciumchlorid,
Natriumdihydrogenphosphat, Magnesiumchlorid usw. Außerdem können Hefe,
Vitamine, Wachstumsbeschleunigungsfaktoren usw. ebenso zu dem Medium
zugegeben werden. Bevorzugt wird der pH des Mediums auf etwa 5 bis
etwa 8 eingestellt.
-
Ein
bevorzugtes Beispiel des Mediums zur Inkubation der Bakterien, die
zu der Gattung Escherichia gehören,
ist M9-Medium, ergänzt
mit Glukose und Casaminosäuren
[Miller, Journal of Experiments in Molecular Genetics, 431–433, Cold
Spring Harbor Laboratory, New York, 1972]. Wenn notwendig, kam eine
Chemikalie wie 3β-Indolylacrylsäure zu dem
Medium zugegeben werden, wodurch der Promotor effizient aktiviert wird.
-
Wenn
die Bakterien, die zu der Gattung Escherichia gehören, als
der Wirt verwendet werden, wird die Transformante normalerweise
bei etwa 15 °C
bis etwa 43 °C
für etwa
3 Stunden bis etwa 24 Stunden kultiviert. Wenn notwendig, kann die
Kultur belüftet
oder gerührt
werden.
-
Wenn
die Bakterien, die zu der Gattung Bacillus gehören, als der Wirt verwendet
werden, wird die Transformante im allgemeinen bei etwa 30 °C bis etwa
40 °C für etwa 6
Stunden bis etwa 24 Stunden kultiviert. Wenn notwendig, kann die
Kultur belüftet
oder gerührt
werden.
-
Wenn
Hefe als Wirt verwendet wird, wird die Transformante beispielsweise
in Burkholder-Minimalmedium
[Bostian, K. L. et al., (Proc. Natl. Acad. Sci. U.S.A.,) 77, 4505
(1980)] oder in SD-Medium, ergänzt
mit 0,5 % Casaminosäuren,
kultiviert [Bitter, G. A. et al., (Proc. Natl. Acad. Sci. U.S.A.,)
81, 5330 (1984)]. Bevorzugt wird der pH des Mediums auf etwa 5 bis
etwa 8 eingestellt. Im allgemeinen wird die Transformante bei etwa
20 °C bis
etwa 35 °C
für etwa
24 Stunden bis etwa 72 Stunden kultiviert. Wenn notwendig, kann
die Kultur belüftet
oder gerührt
werden.
-
Wenn
Insektenzellen oder Insekten als Wirt verwendet werden, wird die
Transformante in Grace-Insektenmedium kultiviert (Grace, T. C. C.,
Nature, 195, 788 (1962)), zu dem ein geeignetes Additiv, wie immobilisiertes
10%iges Rinderserum, zugegeben wird. Bevorzugt wird der pH des Mediums
auf etwa 6,2 bis etwa 6,4 eingestellt. Normalerweise wird die Transformante
bei etwa 27 °C
für etwa
3 Tage bis etwa 5 Tage kultiviert, und wenn notwendig, kann die
Kultur belüftet
oder gerührt
werden.
-
Wenn
Tierzellen als Wirt eingesetzt werden, wird die Transformante in
beispielsweise MEM-Medium, umfassend
etwa 5 % bis etwa 20 % fetales Rinderserum, [Science, 122, 501 (1952)],
DMEM-Medium [Virology, 8, 396 (1959)], RPMI-1640-Medium [The Journal
of the American Medical Association, 199, 519 (1967)], 199-Medium
[Proceeding of the Society for the Biological Medicine, 73, 1 (1950)],
usw. kultiviert. Bevorzugt wird der pH des Mediums auf etwa 6 bis
etwa 8 eingestellt. Die Transformante wird normalerweise bei etwa
30 °C bis
etwa 40 °C
für etwa
15 Stunden bis etwa 60 Stunden kultiviert, und wenn notwendig, kann
die Kultur belüftet
oder gerührt
werden.
-
Wie
oben beschrieben, kann das Peptid/Protein in der Zelle, Zellmembran
oder der Außenseite
der Transformante usw. hergestellt werden.
-
Das
Peptid/Protein kann von der oben beschriebenen Kultur durch die
folgenden Verfahren abgetrennt und gereinigt werden.
-
Wenn
das Peptid/Protein aus der Kultur oder Zellen extrahiert wird, werden
nach der Kultivierung die Transformanten oder Zellen durch ein öffentlich
bekanntes Verfahren gesammelt und in einem geeigneten Puffer suspendiert.
Die Transformaten oder Zellen werden dann durch öffentlich bekannte Verfahren,
wie Ultraschallbehandlung, eine Behandlung mit Lysozym und/oder
Gefrier-Tau-Kreislauf, gefolgt von Zentrifugation, Filtration usw.
gespalten. So kann das rohe Extrakt des Proteins erhalten werden.
Der Puffer, der für
die Verfahrensweisen verwendet wird, kann einen Proteinmodifikator,
wie Harnstoff oder Guanidinhydrochlorid, oder ein oberflächenaktives
Mittel, wie Triton X-100TM, usw. umfassen.
Wenn das Peptid/Protein in der Kultur sekretiert wird, kann nach
der Beendigung der Kultivierung der Überstand von den Transformanten
oder Zellen abgetrennt werden, um den Überstand durch ein öffentlich
bekanntes Verfahren zu sammeln.
-
Das
Peptid/Protein, das in dem so erhaltenen Kulturüberstand oder Extrakt enthalten
ist, kann durch geeignetes Kombinieren der öffentlich bekannten Verfahren
zur Trennung und Reinigung gereinigt werden. Diese öffentlich
bekannten Verfahren zur Trennung und Reinigung umfassen ein Verfahren,
das den Unterschied in der Löslichkeit
nutzt, wie Aussalzen, Lösungsmittelausfällung usw.;
ein Verfahren, das hauptsächlich den
Unterschied im Molekulargewicht nutzt, wie Dialyse, Ultrafiltration,
Gelfiltration, SDS-Polyacrylamidgelelektrophorese usw.; ein Verfahren,
das den Unterschied in der elektrischen Ladung nutzt, wie Ionenaustauschchromatographie
usw.; ein Verfahren, das den Unterschied in der spezifischen Affinität nutzt,
wie Affinitätschromatographie
usw.; ein Verfahren, das den Unterschied in der Hydrophobie nutzt,
wie Umkehrphasen-Hochleistungsflüssigchromatographie
usw.; ein Verfahren, das den Unterschied im isoelektrischen Punkt
nutzt, wie isoelektrische Fokussierungselektrophorese; und dergleichen.
-
Wenn
das so erhaltene Peptid/Protein in freier Form vorliegt, kann es
in das Salz durch öffentlich
bekannte Verfahren oder Modifikationen davon umgewandelt werden.
Wenn andererseits das Protein in Form eines Salzes erhalten wird,
kann es in die freie Form oder in die Form eines anderen Salzes
durch öffentlich
bekannte Verfahren oder Modifikationen davon umgewandelt werden.
-
Das
Peptid/Protein, das durch die Rekombinate hergestellt wird, kann
vor oder nach der Reinigung mit einem geeigneten Protein-modifizierenden
Enzym behandelt werden, so daß das
Protein geeignet modifiziert oder teilweise entfernt werden kann.
Beispiele des Proteinmodifizierenden Enzyms umfassen Trypsin, Chymotrypsin,
Arginylendopeptidase, Proteinkinase, Glykosidase oder dergleichen.
-
Die
Aktivität
des so hergestellten Peptids der Erfindung kann durch einen Bindungstest
an eine markierte Form des Peptids der Erfindung und des gegenwärtigen Proteins,
durch einen Enzymimmunoassay unter Verwendung eines speziellen Antikörpers oder
dergleichen bestimmt werden.
-
Die
Aktivität
des hergestellten gegenwärtigen
Proteins der Erfindung kann durch den Bindungstest an eine markierte
Form des Peptids der Erfindung, durch einen Enzymimmunoassay unter
Verwendung eines speziellen Antikörpers oder dergleichen bestimmt
werden.
-
Die
Antikörper
für das
Peptid der Erfindung, das gegenwärtige
Protein der Erfindung, seine Teilpeptide oder Salze davon können irgendwelche
polyklonalen Antikörper
und monoklonalen Antikörper
sein, so lange wie sie das Peptid der Erfindung, das gegenwärtige Protein
der Erfindung, seine Teilpeptide oder Salze davon erkennen können.
-
Die
Antikörper
für das
Peptid der Erfindung, das gegenwärtige
Protein der Erfindung, seine Teilpeptide oder Salze davon (hierin
nachstehend manchmal nur als das Peptid/Protein usw. der Erfindung
bezeichnet) können
durch öffentlich
bekannte Verfahren zur Herstellung von Antikörpern oder Antisera unter Verwendung des
Peptids/Proteins usw. der Erfindung als Antigene hergestellt werden.
-
[Herstellung des monoklonalen
Antikörpers]
-
(a) Herstellung von Zellen,
die den monoklonalen Antikörper
produzieren
-
Das
Peptid/Protein usw. wird den Säugern
entweder allein oder zusammen mit Trägern oder Verdünnungsmitteln
an der Stelle, wo die Produktion des Antikörpers durch die Verabreichung
möglich
ist, verabreicht. Um die Antikörperproduktivität bei der
Verabreichung zu verstärken,
können
komplettes Freund-Adjuvans oder inkomplettes Freund-Adjuvans verabreicht
werden. Die Verabreichung wird normalerweise einmal aller zwei bis
sechs Wochen und zwei- bis zehnmal insgesamt durchgeführt. Beispiele
der verwendbaren Säuger
sind Affen, Kaninchen, Hunde, Meerschweinchen, Mäuse, Ratten, Schafe und Ziegen,
wobei die Verwendung von Mäusen
und Ratten bevorzugt ist.
-
Bei
der Herstellung von Zellen, die den monoklonalen Antikörper produzieren,
wird ein Warmbluttier, beispielsweise Mäuse, die mit einem Antigen
immunisiert sind, wobei auf den Antikörpertiter geachtet wird, ausgewählt, dann
wird die Milz oder der Lymphknoten nach zwei bis fünf Tagen
nach der letzten Immunisierung gesammelt und die darin enthaltenden
Antikörper-produzierenden
Zellen werden mit Myelomzellen fusioniert, wodurch Hybridome, die
den monoklonalen Antikörper
produzieren, erhalten wurden. Die Messung des Antikör pertiters
in Antisera kann beispielsweise durch Umsetzen eines markierten
Peptids/Proteins, das später
beschrieben wird, mit dem Antiserum, gefolgt von Analysieren der
Bindungsaktivität
des Markierungsmittels, das an den Antikörper bindet, durchgeführt werden.
Die Fusionierung kann beispielsweise durch das bekannte Verfahren
von Koehler und Milstein [Nature, 256, 495, (1975)] durchgeführt werden.
Beispiele des Fusionsbeschleunigers sind Polyethylenglykol (PEG),
Sendai-Virus usw., wobei PEG bevorzugt eingesetzt wird.
-
Beispiele
der Myelomzellen sind gesammeltes NS-1, P3U1, SP2/0 usw. Insbesondere
wird P3U1 bevorzugt eingesetzt. Ein bevorzugtes Verhältnis der
Zahl der verwendeten Antikörperproduzierenden
Zellen (Milzzellen) zu der Zahl der Myelomzellen liegt innerhalb
eines Bereiches von ungefähr
1 : 1 bis 20 : 1. Wenn PEG (bevorzugt PEG 1000 bis PEG 6000) in
einer Konzentration von ungefähr
10 bis 80 % zugegeben wird, gefolgt von Inkubieren bei etwa 20 bis
40 °C, bevorzugt
bei 30 bis 37 °C
für etwa
1 bis 10 Minuten, kann eine effiziente Zellfusion durchgeführt werden.
-
Verschiedene
Verfahren können
zum Screenen eines Hybridoms, das den monoklonalen Antikörper produziert,
verwendet werden. Beispiele von solchen Verfahren umfassen ein Verfahren,
umfassend das Zugeben des Überstandes
von Hybridom zu einer festen Phase (beispielsweise Mikroplatte),
an der das Peptid/Protein usw. der Erfindung als ein Antigen direkt
oder zusammen mit einem Träger
adsorbiert ist, Zugeben eines Anti-Immunoglobulin-Antikörpers (wenn
Mauszellen für
die Zellfusion verwendet werden, wird Anti-Maus-Immunoglobulin-Antikörper verwendet), markiert mit
einer radioaktiven Substanz oder einem Enzym oder Protein A, und
Detektieren des monoklonalen Antikörpers, der an die feste Phase
gebunden ist, und ein Verfahren, umfassend das Zugeben des Uberstandes
von Hybridom zu einer festen Phase, an der ein Anti-Immunoglobulin-Antikörper oder
Protein A adsorbiert ist, Zugeben des Peptids/Proteins usw., markiert
mit einer radioaktiven Substanz oder einem Enzym, und Detektieren
des monoklonalen Antikörpers,
der an die feste Phase gebunden ist.
-
Der
monoklonale Antikörper
kann gemäß öffentlich
bekannten Verfahren oder ihren Modifikationen ausgewählt werden.
Im allgemeinen kann die Selektion in einem Medium für Tierzellen,
ergänzt
mit HAT (Hypoxanthin, Aminopterin und Thymidin), bewirkt werden.
Jedes Selektions- und Wachstumsmedium kann eingesetzt werden, insofern
das Hybridom dort wachsen kann. Beispielsweise kann RPMI-1640-Medium,
umfassend 1 % bis 20 %, bevorzugt 10 % bis 20 % fetales Rinderserum,
GIT-Medium (Wako Pure Chemical Industries, Ltd.), umfassend 1 %
bis 10 % fetales Rinderserum, ein serumfreies Medium zur Kultivierung
eines Hybridoms (SFM-101, Nissui Seiyaku Co., Ltd.) und dergleichen
für das
Selektions- und Wachstumsmedium verwendet werden. Die Kultivierung
wird im allgemeinen bei 20 °C
bis 40 °C,
bevorzugt bei 37 °C,
für etwa
5 Tage bis etwa 3 Wochen, bevorzugt 1 bis 2 Wochen, normalerweise
in 5 % CO2 durchgeführt. Der Antikörpertiter des
Kulturüberstandes
eines Hybridoms kann wie in dem oben beschriebenen Assay für den Antikörpertiter
in Antisera bestimmt werden.
-
(b) Reinigung des monoklonalen
Antikörpers
-
Trennung
und Reinigung eines monoklonalen Antikörpers kann durch konventionelle
Verfahren durchgeführt
werden, wie Trennungs- und Reinigungsverfahren des polyklonalen
Antikörpers,
wie Trennung und Reinigung von Immunoglobulinen [beispielsweise
Aussalzen, Alkoholausfällung,
isoelektrische Punktausfällung,
Elektrophorese, Adsorption und Desorption mit Ionenaustauschern
(z.B. DEAE), Ultrazentrifugation, Gelfiltration oder ein spezifisches
Reinigungsverfahren, welches nur das Sammeln eines Antikörpers mit
einem aktivierten Adsorptionsmittel, wie eine Antigen-Bindungsfestphase,
Protein A oder Protein G, und Dissoziieren der Bindung, um den Antikörper zu
erhalten, umfaßt].
-
[Herstellung des polyklonalen
Antikörpers]
-
Der
polyklonale Antikörper
der vorliegenden Erfindung kann durch öffentlich bekannte Verfahren
oder Modifikationen davon hergestellt werden. Beispielsweise wird
ein Säuger
mit einem Immunogen (das Peptid/Protein usw. der Erfindung als ein
Antigen) per se immunisiert, oder ein Komplex aus Immunogen und
einem Trägerprotein
wird gebildet und ein Warmbluttier wird mit dem Komplex in einer ähnlichen
Weist zu dem oben beschriebenen Verfahren für die Herstellung von monoklonalen
Antikörpern
immunisiert. Das Produkt, das den Antikörper für das Peptid/Protein usw. der
Erfindung umfaßt,
wird aus dem immunisierten Tier gesammelt, gefolgt von Trennung
und Reinigung des Antikörpers.
-
In
dem Komplex aus Immunogen und Trägerprotein,
der verwendet wird, um einen Säuger
zu immunisieren, kann der Typ des Trägerproteins und das Mischverhältnis von
Träger
zu Hap ten jeder Typ und jedes Verhältnis sein, so lange wie der
Antikörper
effizient für
das Hapten, das durch Vernetzen mit dem Träger immunisiert wird, produziert
wird. Beispielsweise wird Rinderserumalbumin, Rinderthyroglobulin
oder Schlüssellochlimpethemocyanin
usw. mit dem Hapten in einem Träger-zu-Hapten-Gewichtsverhältnis von
ungefähr
0,1 bis 20, bevorzugt etwa 1 bis etwa 5 verknüpft.
-
Eine
Vielzahl von Kondensationsmitteln kann zum Verknüpfen von Träger mit Hapten verwendet werden.
Glutaraldehyd-, Carbodiimid-, Maleimid-aktivierter Ester und aktivierte
Esterreagenzien, umfassend eine Thiolgruppe oder Dithiopyridylgruppe,
werden zum Verknüpfen
verwendet.
-
Das
Kondensationsprodukt wird den Warmbluttieren entweder allein oder
zusammen mit Trägern
oder Verdünnungsmitteln
an der Stelle, die den Antikörper
durch Verabreichung produzieren kann, verabreicht. Um die Antikörperproduktivität bei der
Verabreichung zu verstärken,
kann komplettes Freund-Adjuvans oder inkomplettes Freund-Adjuvans
verabreicht werden. Die Verabreichung wird normalerweise aller 2
bis 6 Wochen und 3 bis 10 Mal insgesamt durchgeführt.
-
Der
polyklonale Antikörper
kann aus Blut, Aszites usw., bevorzugt aus dem Blut des Säugers, der durch
das oben beschriebene Verfahren immunisiert wird, gesammelt werden.
-
Der
Titer des polyklonalen Antikörpers
in Antiserum kann durch dieselbe Verfahrensweise wie die zur Bestimmung
des oben beschriebenen Serumantikörpertiters analysiert werden.
Die Trennung und Reinigung des polyklonalen Antikörpers kann
durchgeführt
werden, indem dem Verfahren zur Trennung und Reinigung von Immunoglobulinen,
das wie bei der Trennung und Reinigung von hierin zuvor beschriebenen
monoklonalen Antikörpern
durchgeführt
wird, gefolgt wird.
-
Das
Peptid der Erfindung, die DNA, die das Peptid der Erfindung kodiert
(hierin nachstehend manchmal nur als die erfindungsgemäße DNA bezeichnet),
und der Antikörper
für das
Peptid der Erfindung (hierin nachstehend manchmal nur als der erfindungsgemäße Antikörper bezeichnet)
sind nützlich
zum Implementieren (1) von Mitteln für die Vorbeugung/Behandlung
von verschiedenen Krankheiten, die mit dem Protein der Erfindung
verbunden sind; (2) Screenen einer Verbindung oder ihres Salzes,
die die Bindungseigenschaft des Peptids der Erfindung an das Protein
der Erfindung verändert;
(3) Quantifikation des Peptids der Erfindung oder seines Salzes;
(4) Gendiagnose; (5) Arzneimittel, umfassend die Antisense-DNA;
(6) Arzneimittel, umfassend den Antikörper der Erfindung; (7) Präparieren
nicht-menschlicher
Tiere, die die erfindungsgemäße DNA tragen;
(8) Arzneimitteldesign, basierend auf dem Vergleich zwischen Liganden
und Rezeptoren, die strukturell analog sind, usw.
-
Das
Peptid der Erfindung, die erfindungsgemäße DNA und der erfindungsgemäße Antikörper werden speziell
in bezug auf ihre Anwendungen nachstehend beschrieben.
-
(1) Therapeutika/Präventivmittel
für Krankheiten,
mit denen das Peptid der Erfindung verbunden ist
-
Wie
hierin nachstehend beschrieben wird, ist klar geworden, daß das Peptid
der Erfindung ein Ligand für
das Protein der Erfindung ist (G-Protein-gekoppelter Rezeptor),
da das Peptid der Erfindung als ein humoraler Faktor in vivo vorliegt
und das Protein der Erfindung aktiviert, um die intrazelluläre Ca2+-Ionenkonzentration in der Zelle zu erhöhen, in
der das Protein der Erfindung exprimiert worden ist.
-
Ebenso
wird erkannt, daß das
Peptid der Erfindung etwa 56 % Homologie zu Schlangengift Mamba Intestinal
Toxin 1 (manchmal als MIT1 abgekürzt;
SEQ ID NR. 34; Toxicon, 28, 847–856,
1990; FEBS Letters, 461, 183–188,
1999) auf einem Aminosäureniveau
aufweist.
-
Es
wurde berichtet, daß MIT1
die Kontraktion im Krummdarm oder distalen Dickdarm oder Relaxation im
proximalen Dickdarm induziert, und sein Grad war so wirksam wie
vergleichbar mit 40 mM Kaliumchlorid (FEBS Letters, 461, 183–188, 1999).
Jedoch wurde die Stelle oder der Mechanismus seiner Wirkung nicht
gelöst.
Die betreffenden Erfinder klärten,
daß die
Wirkung von MIT1 ebenso durch das Protein der Erfindung vermittelt
wurde.
-
Im
Hinblick auf das Vorhergehende weist das Peptid der Erfindung eine
Aktivität
zum Induzieren der Kontraktion oder Relaxation der gesamten Darmtrakte
auf, um die Darmtraktmotilität
(Verdauungsaktivität) usw.
zu kontrollieren (z.B. später
beschriebenes BEISPIEL 11). Wenn daher die DNA usw. der Erfindung
fehlt oder wenn ihr Expressionsniveau abnormal verringert wird,
entwickeln sich verschiedene Krankheiten, wie Verdauungsstörungen (z.B.
Enteritis, Verstopfung, Malabsorptionssyndrom usw.).
-
Folglich
kann das Peptid der Erfindung oder die DNA der Erfindung als Arzneimittel
für die
Behandlung oder Vorbeugung usw. von verschiedenen Krankheiten verwendet
werden, einschließlich
Verdauungsstörungen
(z.B. Enteritis, Verstopfung, Malabsorptionssyndrome usw.) oder
dergleichen.
-
Wenn
ein Patient ein verringertes Niveau an dem Peptid der Erfindung
in seinem Körper
aufweist oder es ihm daran dort mangelt, so daß die Signaltransduktion nicht
vollständig
oder normal in den Zellen, in denen das Protein der Erfindung exprimiert
wird, ausgeübt
wird, kann das Peptid der Erfindung seine Rolle ausreichend oder
geeignet für
den Patient bereitstellen, (a) durch Verabreichen der erfindungsgemäßen DNA
an den Patienten, um das Peptid der Erfindung in dem Körper zu
exprimieren, (b) durch. Einführen
der erfindungsgemäßen DNA
in eine Zelle, die das Peptid der Erfindung exprimiert, und dann
Transplantieren der Zellen in den Patienten, oder (c) durch Verabreichen
des Peptids der Erfindung an den Patienten, oder dergleichen.
-
Wenn
die erfindungsgemäße DNA als
die oben beschriebenen Präventivmittel/Therapeutika
verwendet werden, wird die DNA selbst an den Menschen oder ein anderes
Warmbluttier verabreicht; oder die DNA wird in einen geeigneten
Vektor, wie Retrovirusvektor, Adenovirusvektor, Adenovirus-assoziierter
Virusvektor usw., eingeführt
und dann an den Menschen oder ein anderes Warmbluttier in einer
konventionellen Weise verabreicht. Die erfindungsgemäße DNA kann
ebenso als eine intakte DNA verabreicht werden; oder die DNA kann
zu einer pharmazeutische Zusammensetzung mit physiologisch akzeptablen
Trägern,
wie Hilfsmitteln usw., hergestellt werden, um ihre Aufnahme zu beschleunigen,
und die Zusammensetzung kann durch Genkanone oder durch einen Katheter,
wie einen Katheter mit einem Hydrogel, verabreicht werden.
-
Wenn
das Peptid der Erfindung als die zuvor genannten Therapeutika/Präventivmittel
verwendet wird, wird das Peptid vorteilhafterweise auf einem Reinheitsniveau
von mindestens 90 %, bevorzugt mindestens 95 %, stärker bevorzugt
mindestens 98 % und am stärksten
bevorzugt mindestens 99 % verwendet.
-
Das
Peptid der Erfindung kann oral, beispielsweise in Form von Tabletten,
die nach Bedarf zuckerüberzogen
sein können,
Kapseln, Elixieren, Mikrokapseln usw., oder parenteral in Form von.
Injektionspräparaten,
wie einer sterilen Lösung
und einer Suspension in Wasser oder mit einer anderen pharmazeutisch
akzeptablen Flüssigkeit,
verwendet werden. Diese Präparate
können
durch Mischen des Peptids der Erfindung mit einem physiologisch
akzeptablen bekannten Träger,
einem Aromastoff einem Trägerstoff,
einem Vehikel, einem Antiseptikum, einem Stabilisator, einem Bindemittel
usw. in einer Einheitsdosierungsform hergestellt werden, die in
einer allgemein akzeptierten Weise erforderlich ist, die auf die
Herstellung von pharmazeutischen Präparaten angewendet wird. Der
Wirkstoff in dem Präparat
wird in einer solchen Dosis kontrolliert, daß eine geeignete Dosis innerhalb
des angegebenen spezifizierten Bereiches erhalten wird.
-
Additive,
die mit Tabletten, Kapseln usw. mischbar sind, umfassen ein Bindmittel,
wie Gelatine, Maisstärke,
Tragant und Gummiarabikum, einen Trägerstoff, wie kristalline Cellulose,
ein Quellungsmittel, wie Maisstärke,
Gelatine, Alginsäure
usw., ein Schmiermittel, wie Magnesiumstearat, ein Süßungsstoff,
wie Saccharose, Lactose und Saccharin, und einen Geschmacksstoff,
wie Pfefferminze, Akamonoöl
oder Kirsche usw. Wenn die Einheitsdosierungen in der Form von Kapseln
vorliegen, können
flüssige
Träger
wie Öle
und Fette außerdem
zusammen mit den oben beschriebenen Additiven verwendet werden.
Eine sterile Zusammensetzung zur Injektion kann gemäß einer
konventionellen Weise formuliert werden, die verwendet wird, um
pharmazeutische Zusammensetzungen herzustellen, beispielsweise durch
Lösen oder
Suspendieren der Wirkstoffe in einem Vehikel, wie Wasser zur Injektion
mit einem natürlich
vorkommenden Pflanzenöl,
wie Sesamöl
und Kokosnußöl usw.,
um die pharmazeutische Zusammensetzung herzustellen.
-
Beispiele
eines wässerigen
Mediums zur Injektion umfassen physiologische Kochsalzlösung und
eine isotonische Lösung,
die Glukose und andere Hilfsmittel enthält (z.B. D-Sorbitol, D-Mannitol, Natriumchlorid usw.),
und kann in Kombination mit einem geeigneten Auflösungshilfsmittel,
wie einem Alkohol (z.B. Ethanol oder dergleichen), einem Polyalkohol
(z.B. Propylenglykol und Polyethylenglykol), einem nicht-ionischen
oberflächenaktiven
Mittel (z.B. Polysorbate 80TM, HCO-50 usw.)
oder dergleichen verwendet werden. Beispiele des öligen Mediums
umfassen Sesamöl
und Sojabohnenöl,
das ebenso in Kombination mit einem Auflösungshilfsmittel wie Benzylbenzoat
und Benzylalkohol verwendet werden kann. Sie können außerdem mit einem Puffer (z.B.
Phosphatpuffer, Natriumacetatpuffer usw.), einem Desinfektionsmittel
(z.B. Benzalkoniumchlorid, Prokainhydrochlorid usw.), einem Stabilisator
(z.B. menschliches Serumalbumin, Polyethylenglykol usw.), einem Konservierungsmittel
(z.B. Benzylalkohol, Phenol usw.), einem Antioxidationsmittel usw.
formuliert werden. Die so hergestellte Flüssigkeit zur Injektion wird
normalerweise in eine geeignete Ampulle gefüllt.
-
Der
Vektor, in den die erfindungsgemäße DNA eingeführt wird,
kann ebenso zu pharmazeutischen Präparaten in einer Weise hergestellt
werden, die den obigen Verfahrensweisen ähnlich ist. Diese Präparate werden
im allgemeinen parenteral verwendet.
-
Da
das so erhaltene pharmazeutische Präparat sicher und gering toxisch
ist, kann das Präparat
an den Säuger
verabreicht werden (z.B. Mensch, Ratte, Maus, Meerschweinchen, Kaninchen,
Schaf, Schwein, Rind, Pferd, Katze, Hund, Affe usw.).
-
Die
Dosis des Peptids der Erfindung variiert in Abhängigkeit der Zielkrankheit,
des Patienten, an den sie verabreicht werden soll, der Verabreichungsweise
usw.; beispielsweise beträgt
bei der oralen Verabreichung des Peptids der Erfindung für die Behandlung
einer Verdauungsstörung
die Dosis normalerweise etwa 1 mg bis etwa 1000 mg, bevorzugt etwa
10 bis etwa 500 mg, und stärker
bevorzugt etwa 10 bis etwa 200 mg pro Tag für Erwachsene (bei 60 kg Körpergewicht).
Bei der parenteralen Verabreichung variiert die Einzeldosis in Abhängigkeit
des Patienten, an den sie verabreicht werden soll, der Zielkrankheit
usw., aber es ist für
die Behandlung einer Verdauungsstörung vorteilhaft, den Wirkstoff
intravenös
bei einer täglichen
Dosis von etwa 1 bis etwa 1000 mg, bevorzugt etwa 1 bis etwa 200
mg, und stärker
bevorzugt etwa 10 bis etwa 100 mg für Erwachsene (bei 60 kg Körpergewicht)
zu verabreichen. Für
andere Tierspezies kann die entsprechende Dosis, auf 60 kg Körpergewicht
umgerechnet, verabreicht werden.
-
(2) Pharmazeutische Zusammensetzung,
umfassend den erfindungsgemäßen Antikörper
-
Der
Antikörper
der Erfindung mit der Wirkung, das Peptid der Erfindung zu neutralisieren,
kann als Präventivmittel/Therapeutika
für Krankheiten
verwendet werden, die mit der Übe rexprimierung
des Peptids der Erfindung verbunden sind (beispielsweise Verdauungsstörungen (z.B.
Enteritis, Durchfall, Verstopfung, Malabsorptionssyndrom usw.)).
-
Das
Therapeutika/Präventivmittel
für die
oben beschriebenen Krankheiten, umfassend den Antikörper der
Erfindung, können
an den Menschen oder ein anderes Warmbluttier (z.B. Ratte, Kaninchen,
Schaf, Schwein, Rind, Katze, Hund, Affe usw.) direkt als ein flüssiges Präparat oder
als eine pharmazeutische Zusammensetzung in einer geeigneten Präparatform
oral oder parenteral verabreicht werden. Die Dosis variiert in Abhängigkeit
des Patienten, an den sie verabreicht werden soll, der Zielkrankheit,
den Zuständen,
der Verabreichungsweise usw.; wenn sie für die Behandlung/Vorbeugung
des erwachsenen Patienten mit beispielsweise Verdauungsstörung verwendet
wird, wird der Antikörper
der Erfindung vorteilhaft an den Patienten durch intravenöse Injektion
verabreicht, normalerweise in einer Einzeldosis von ungefähr 0,01
bis 20 mg/kg Körpergewicht,
bevorzugt etwa 0,1 bis etwa 10 mg/kg Körpergewicht, und stärker bevorzugt
etwa 0,1 bis etwa 5 mg/kg Körpergewicht,
ungefähr
ein- bis fünfmal,
bevorzugt ungefäh
ein- bis dreimal pro Tag. Für
die andere parenterale Verabreichung und orale Verabreichung kann
die entsprechende Dosis verabreicht werden. Wenn die Zustände sehr
ernst sind, kann die Dosis in Abhängigkeit der Zustände erhöht werden.
-
Der
Antikörper
der Erfindung kann direkt als solcher oder in Form einer geeigneten
pharmazeutischen Zusammensetzung verabreicht werden. Die pharmazeutische
Zusammensetzung, die für
die oben beschriebene Verabreichung verwendet wird, enthält einen
pharmakologisch akzeptablen Träger
mit den zuvor genannten Verbindungen oder Salzen davon, ein Verdünnungsmittel
oder Trägerstoff.
Eine solche Zusammensetzung wird in dem Präparat, das zur oralen oder
parenteralen Verabreichung geeignet ist, bereitgestellt.
-
Das
heißt,
Beispiele der Zusammensetzung zur oralen Verabreichung umfassen
feste oder flüssige Präparate,
speziell Tabletten (einschließlich
Dragees und Tabletten in Filmhüllenform),
Pillen, Granulate, pulverförmige
Präparate,
Kapseln (einschließlich
weiche Kapseln), Sirup, Emulsionen, Suspensionen usw. Eine solche
Zusammensetzung wird durch öffentlich
bekannte Verfahren hergestellt und enthält ein Vehikel, ein Verdünnungsmittel
oder einen Trägerstoff
der konventionell in dem Bereich der pharmazeutischen Präparate verwendet
wird. Beispiele des Vehikels oder Trägerstoffes für Tabletten
sind Lactose, Stärke,
Saccharose, Magnesiumstearat usw.
-
Beispiele
der Zusammensetzung zur parenteralen Verabreichung, die verwendet
werden kann, sind Injektionen, Zäpfchen
usw., und die Injektionen umfassen die Form von intravenösen, subkutanen,
transkutanen, intramuskulären
und Tropfinjektionen usw. Diese Injektionen werden durch öffentlich
bekannte Verfahren hergestellt, beispielsweise durch Lösen, Suspendieren
oder Emulgieren des zuvor genannten Antikörpers oder seiner Salze in
einem sterilen wässerigen
oder öligen
flüssigen
Medium. Für
das wässerige
Medium zur Injektion werden beispielsweise physiologische Kochsalzlösung und
isotonische Lösungen,
die Glukose und anderes Hilfsmittel enthalten., usw. verwendet.
Geeignete Auflösungshilfsmittel,
beispielsweise Alkohol (z.B. Ethanol), Polyalkohol (z.B. Propylenglykol
oder Polyethylenglykol), nicht-ionische oberflächenaktive Mittel [z.B. Polysorbat
80, HCO-50 (Polyoxyethylenaddukt (50 mol) von hydriertem Rizinusöl)] können in
Kombination verwendet werden. Für
die ölige
Lösung
werden beispielsweise Sesamöl,
Sojabohnenöl
und dergleichen verwendet, und Auflösungshilfsmittel, wie Benzylbenzoat,
Benzylalkohol usw. können
in Kombination verwendet werden. Die so hergestellte Flüssigkeit
zur Injektion wird normalerweise in eine geeignete Ampulle gefüllt. Das Zäpfchen,
das zur rektalen Verabreichung verwendet wird, wird durch Mischen
des zuvor genannten Antikörpers
oder seiner Salze mit konventionellen Zäpfchengrundlagen hergestellt.
-
Die
oben beschriebene orale oder parenterale pharmazeutische Zusammensetzung
wird vorteilhaft in einer Einheitsdosierungsform hergestellt, die
für die
Dosis des Wirkstoffes geeignet ist. Beispiele einer solchen Einheitsdosierungsform
umfassen Tabletten, Pillen, Kapseln, Injektionen (Ampullen), Zäpfchen usw.
Es ist bevorzugt, daß der
oben beschriebene Antikörper
im allgemeinen in einer Dosis von 5 bis 500 mg pro Einheitsdosierungsform,
5 bis 100 mg speziell für
Injektionen und 10 bis 250 mg für
andere Präparate
enthalten ist.
-
Jede
oben beschriebene Zusammensetzung kann außerdem andere aktive Komponenten
enthalten, wenn die Formulierung mit dem Antikörper keine nachteilige Interaktion
hervorruft.
-
(3) Präparieren von nicht-menschlichen
Tieren, die die erfindungsgemäße DNA tragen.
-
Unter
Verwendung der erfindungsgemäßen DNA
können
nicht-menschliche transgene Tiere, die das Protein usw. der Erfindung
exprimieren, präpariert
werden. Beispiele der nicht- menschlichen
Tiere umfassen Säuger
(beispielsweise Ratten, Mäuse,
Kaninchen, Schafe, Schweine, Rinder, Katzen, Hunde, Affen usw.) (hierein
nachstehend nur als Tiere bezeichnet), wobei Mäuse und Kaninchen besonders
geeignet sind.
-
Um
die erfindungsgemäße DNA auf
das Zieltier zu übertragen,
ist es im allgemeinen vorteilhaft, die DNA in einem Genkonstrukt
zu verwenden, das stromabwärts
eines Promotors ligiert wird, der die DNA in Tierzellen exprimieren
kann. Wenn beispielsweise die erfindungsgemäße DNA, die von Kaninchen abgeleitet
ist, übertragen
wird, wird beispielsweise das Genkonstrukt, in dem die DNA stromabwärts eines
Promotors ligiert wird, der die erfindungsgemäße DNA, abgeleitet von Tieren,
exprimieren kann, umfassend die erfindungsgemäße DNA, die stark zu der Kaninchen-abgeleiteten
DNA homolog ist, in befruchtete Kanincheneizellen mikroinjiziert;
daher kann das DNA-übertragene
Tier, das ein hohes Niveau des Proteins usw. der Erfindung produzieren
kann, hergestellt werden. Beispiele der Promotoren, die nutzbar
sind, umfassen Virus-abgeleitete Promotoren und ubiquitäre Expressionspromotoren,
wie ein Metallothioneinpromotor, aber Promotoren von NGF-Gen und
Enolase, die speziell in dem Gehirn exprimiert werden, werden bevorzugt
verwendet.
-
Die Übertragung
der erfindungsgemäßen DNA
im Stadium der befruchteten Eizelle sichert die Gegenwart der DNA
in allen Keim- und Somazellen in dem produzierten Tier. Die Gegenwart
des Proteins usw. der Erfindung in den Keimzellen in dem DNA-übertragenen
Tier bedeutet, daß alle
Keim- und Somazellen das Protein usw. der Erfindung in allen Nachkommen
des Tiers umfassen. Die Nachkommen des Tieres, die das Gen übernehmen,
umfassen das Protein usw. der Erfindung in allen Keim- und Somazellen.
-
Die
DNA-übertragenen
Tiere der vorliegenden Erfindung können in der konventionellen
Umgebung gehalten und als Tiere, die die DNA nach dem Bestätigen der
stabilen Retention des Gens in den Tieren durch Paarung tragen,
fortgepflanzt werden. Außerdem
führt die
Paarung von männlichen
und weiblichen Tieren, die die Ziel-DNA umfassen, zum Erhalt homozygoter
Tiere mit dem übertragenen
Gen auf beiden homologen Chromosomen. Durch Paarung der männlichen
und weiblichen Homozygoten kann die Fortpflanzung so durchgeführt werden,
daß alle
Nachkommen die DNA umfassen.
-
Da
das Protein usw. der Erfindung stark in den Tieren exprimiert wird,
bei denen die erfindungsgemäße DNA übertragen
worden ist, sind die Tiere zum Screenen von Agonisten oder Antagonisten
zu dem Protein usw. der Erfindung nützlich.
-
Die
Tiere, bei denen die erfindungsgemäße DNA übertragen worden ist, können ebenso
als Zellquellen für
Gewebekultur verwendet werden. Das Protein usw. der Erfindung kann
durch beispielsweise direktes Analysieren der DNA oder RNA in Geweben
aus der Maus, in die die erfindungsgemäße DNA übertragen worden ist, oder
durch Analysieren von Geweben, die das Protein umfassen, welches
aus dem Gen exprimiert wird, analysiert werden. Zellen aus den Geweben,
die das Protein usw. der Erfindung umfassen, werden durch die Standardgewebekulturtechnik
kultiviert. Unter Verwendung dieser Zellen kann beispielsweise die
Funktion von Gewebezellen, wie Zellen, die aus dem Gehirn oder pheripheren
Geweben abgeleitet sind, die im allgemeinen schwer zu kultivieren
sind, untersucht werden. Unter Verwendung dieser Zellen ist es beispielsweise möglich, Pharmazeutika
auszuwählen,
die verschiedene Gewebefunktionen erhöhen. Wenn eine stark exprimierende
Zellinie erhältlich
ist, kann das Protein usw. der Erfindung aus der Zellinie isoliert
und gereinigt werden.
-
In
der Beschreibung und den Zeichnungen werden die Codes von Basen
und Aminosäuren
durch Abkürzungen
gezeigt, und in diesem Fall werden sie gemäß der IUPAC-IUB Commission
on Biochemical Nomenclature oder durch übliche Codes in der Technik
angegeben, Beispiele von denen werden nachstehend gezeigt. Bei Aminosäuren, die
ein optisches Isomer aufweisen können,
liegt die L-Form vor, wenn nicht anders angegeben.
- DNA:
- Deoxyribonukleinsäure
- cDNA:
- komplementäre Deoxyribonukleinsäure
- A:
- Adenin
- T:
- Thymin
- G:
- Guanin
- C:
- Cytosin
- Y:
- Thymin oder Cytosin
- N:
- Thymin, Cytosin, Adenin
oder Guanin
- R:
- Adenin oder Guanin
- M:
- Cytosin oder Adenin
- W:
- Thymin oder Adenin
- S:
- Cytosin oder Guanin
- RNA:
- Ribonukleinsäure
- mRNA:
- Messenger-Ribonukleinsäure
- dATP:
- Deoxyadenosintriphosphat
- dTTP:
- Deoxythymidintriphosphat
- dGTP:
- Deoxyguanosintriphosphat
- dCTP:
- Deoxycytidintriphosphat
- ATP:
- Adenosintriphosphat
- Gly oder G:
- Glycin
- Ala oder A:
- Alanin
- Val oder V:
- Valin
- Leu oder L:
- Leucin
- Ile oder I:
- Isoleucin
- Ser oder S:
- Serin
- Thr oder T:
- Threonin
- Cys oder C:
- Cystein
- Met oder M:
- Methionin
- Glu oder E:
- Glutaminsäure
- Asp oder D:
- Asparaginsäure
- Lys oder K:
- Lysin
- Arg oder R:
- Arginin
- His oder H:
- Histidin
- Phe oder F:
- Phenylalanin
- Tyr oder Y:
- Tyrosin
- Trp oder W:
- Tryptophan
- Pro oder P:
- Prolin
- Asn oder N:
- Asparagin
- Gln oder Q:
- Glutamin
- pGlu:
- Pyroglutaminsäure
- Xaa:
- nicht identifizierter
Aminosäurerest
-
Ebenso
werden die Substituenten, Schutzgruppen, Reagenzien usw., die häufig in
der Beschreibung benutzt werden, durch die folgenden Symbole dargestellt.
- Me:
- Methylgruppe
- Et:
- Ethylgruppe
- Bu:
- Butylgruppe
- Ph:
- Phenylgruppe
- TC:
- Thiazolidin-4(R)-Carboxamidgruppe
- Bom:
- Benzyloxymethyl
- Bzl:
- Benzyl
- Z:
- Benzyloxycarbonyl
- Br-Z:
- 2-Brombenzyloxycarbonyl
- Cl-Z:
- 2-Chlorbenzyloxycarbonyl
- Cl2Bzl:
- 2,6-Dichlorbenzyl
- Boc:
- t-Butyloxycarbonyl
- HOBt:
- 1-Hydroxybenztriazol
- HOOBt:
- 3,4-Dihydro-3-hydroxy-4-oxo-1,2,3-benzotriazin
- PAM:
- Phenylacetamidomethyl
- Tos:
- p-Toluolsulfonyl
- Fmoc:
- N-9-Fluorenylmethoxycarbonyl
- DNP:
- Dinitrophenyl
- Bum:
- tert-Butoxymethyl
- Trt:
- Trityl
- Bom:
- Benzyloxymethyl
- Z:
- Benzyloxycarbonyl
- MeBzl:
- 4-Methylbenzyl
- DCC:
- N,N'-Dicyclohexylcarbodiimid
- HONB:
- 1-Hydroxy-5-norbornen-2,3-dicarboxyimid
- NMP:
- N-Methylpyrrolidon
- HONB:
- N-Hydroxy-5-norbornen-2,3-dicarboxyimid
- NMP:
- N-Methylpyrrolidon
- TFA:
- Trifluoressigsäure
- CHAPS:
- 3-[(3-Cholamidopropyl)dimethylammonio]-1-propansulfonat
- PMSF:
- Phenylmethylsulfonylfluorid
- GDP:
- Guanosin-5'-diphosphat
- Fura-2AM:
- Pentacetoxymethyl-1-[6-amino-2-(5-carboxy-2-oxazolyl)-5-benzofuranyloxy]-2-(2-amino-5-methylphenoxyl)-ethan-N,N,N',N'-tetraacetat
- Fluo-3AM:
- Pentacetoxymethyl-1-[2-amino-5-(2,7-dichlor-6-hydroxy-3-oxy-9-xanthenyl)phenoxy]-2-(2-amino-5-methylphenoxy)-ethan-N,N,N',N'-tetraacetat
- HEPES:
- 2-[4-(2-Hydroxyethyl)-1-piperazinyl]ethansulfonsäure
- EDTA:
- Ethylendiamintetraessigsäure
- SDS:
- Natriumdodecylsulfat
- BSA:
- Rinderserumalbumin
- HBSS:
- Hanks-Mineralsalzmedium
- EIA:
- Enzymimmunoassay
-
Die
Sequenzidentifikationsnummern in dem Sequenzprotokoll der Beschreibung
geben die folgenden Sequenzen an.
-
[SEQ ID NR. 1]
-
Diese
zeigt die Aminosäuresequenz
des Proteins der Erfindung, abgeleitet von menschlichem Gehirn.
-
[SEQ ID NR. 2]
-
Diese
zeigt die Basensequenz der DNA, die das Protein der Erfindung kodiert,
abgeleitet von menschlichem Gehirn, enthaltend die Aminosäuresequenz,
die durch SEQ ID NR. 1 dargestellt ist (ZAQC).
-
[SEQ ID NR. 3]
-
Diese
zeigt die Basensequenz der DNA, die das Protein der Erfindung kodiert,
abgeleitet von menschlichem Gehirn, enthaltend die Aminosäuresequenz,
die durch SEQ ID NR. 1 dargestellt ist (ZAQT).
-
[SEQ ID NR. 4]
-
Diese
zeigt die Basensequenz des Primers 1, verwendet in dem später beschriebenen
BEISPIEL 1.
-
[SEQ ID NR. 5]
-
Diese
zeigt die Basensequenz des Primers 2, verwendet in dem später beschriebenen
BEISPIEL 1.
-
[SEQ ID NR. 6]
-
Diese
zeigt die Basensequenz des Primers 3, verwendet in dem später beschriebenen
BEISPIEL 2.
-
[SEQ ID NR. 7]
-
Diese
zeigt die Basensequenz des Primers 4, verwendet in dem später beschriebenen
BEISPIEL 2.
-
[SEQ ID NR. 8]
-
Diese
zeigt die Basensequenz der ZAQ-Sonde, verwendet in dem später beschriebenen
BEISPIEL 2.
-
[SEQ ID NR. 9]
-
Diese
zeigt die Basensequenz des Primers ZAQC Sal, verwendet in dem später beschriebenen
BEISPIEL 2.
-
[SEQ ID NR. 10]
-
Diese
zeigt die Basensequenz des Primers ZAQC Spe, verwendet in dem später beschriebenen
BEISPIEL 2.
-
[SEQ ID NR. 11]
-
Diese
zeigt die N-terminale Aminosäuresequenz
von ZAQ-aktiviertem Peptid, gereinigt in dem später beschriebenen BEISPIEL
3 (3-8).
-
[SEQ ID NR. 12]
-
Diese
zeigt die Basensequenz des Primers ZF1, verwendet in dem später beschriebenen
BEISPIEL 4.
-
[SEQ ID NR. 13]
-
Diese
zeigt die Basensequenz des Primers ZF2, verwendet in dem später beschriebenen
BEISPIEL 4.
-
[SEQ ID NR. 14]
-
Diese
zeigt die Basensequenz des Primers ZF3, verwendet in dem später beschriebenen
BEISPIEL 4.
-
[SEQ ID NR. 15]
-
Diese
zeigt die 3'-terminate
Basensequenz der DNA, die das menschentypische ZAQ-Ligandenpeptid kodiert,
das in dem später
beschriebenen BEISPIEL 4 erworben wurde.
-
[SEQ ID NR. 16]
-
Diese
zeigt die Basensequenz des Primers ZAQL-CF, verwendet in dem später beschriebenen
BEISPIEL 4.
-
[SEQ ID NR. 17]
-
Diese
zeigt die Basensequenz des Primers ZAQAL-XR1, verwendet in dem später beschriebenen BEISPIEL
4.
-
[SEQ ID NR. 18]
-
Diese
zeigt die Basensequenz des DNA-Fragmentes, erhalten in dem später beschriebenen
BEISPIEL 4.
-
[SEQ ID NR. 19]
-
Diese
zeigt die Basensequenz des DNA-Fragmentes, erhalten in dem später beschriebenen
BEISPIEL 4.
-
[SEQ ID NR. 20]
-
Diese
zeigt die Aminosäuresequenz
des menschentypischen reifen ZAQ-Ligandenpeptids.
-
[SEQ ID NR. 2l]
-
Diese
zeigt die Aminosäuresequenz
des menschentypischen reifen ZAQ-Ligandenpeptids.
-
[SEQ ID NR. 22]
-
Diese
zeigt die Aminosäuresequenz
des menschentypischen ZAQ-Ligandenpräkursorpeptids.
-
[SEQ ID NR. 23]
-
Diese
zeigt die Aminosäuresequenz
des menschentypischen ZAQ-Ligandenpräkursorpeptids.
-
[SEQ ID NR. 24]
-
Diese
zeigt die Basensequenz der DNA, die die DNA enthält, die das menschentypische
ZAQ-Ligandenpräkursorpeptid
kodiert, das durch SEQ ID NR. 28 dargestellt ist.
-
[SEQ ID NR. 25]
-
Diese
zeigt die Basensequenz der DNA, die die DNA enthält, die das menschentypische
ZAQ-Ligandenpräkursorpeptid
kodiert, das durch SEQ ID NR. 29 dargestellt ist.
-
[SEQ ID NR. 26]
-
Diese
zeigt die Basensequenz der DNA, die das menschentypische reife ZAQ-Ligandenpeptid
kodiert, das durch SEQ ID NR. 20 dargestellt ist.
-
[SEQ ID NR. 27]
-
Diese
zeigt die Basensequenz der DNA, die das menschentypische reife ZAQ-Ligandenpeptid
kodiert, das durch SEQ ID NR. 21 dargestellt ist.
-
[SEQ ID NR. 28]
-
Diese
zeigt die Basensequenz der DNA, die das menschentypische ZAQ-Ligandenpräkursorpeptid
kodiert, das durch SEQ ID NR. 22 dargestellt ist.
-
[SEQ ID NR. 29]
-
Diese
zeigt die Basensequenz der DNA, die das menschentypische ZAQ-Ligandenpräkursorpeptid
kodiert, das durch SEQ ID NR. 23 dargestellt ist.
-
[SEQ ID NR. 30]
-
Diese
zeigt die Basensequenz des DNA-Fragmentes, erhalten in dem später beschriebenen
BEISPIEL 5 (5-1).
-
[SEQ ID NR. 31]
-
Diese
zeigt die N-terminale Aminosäuresequenz
von menschentypischem ZAQ-Ligandenpeptid, analysiert in dem später beschriebenen
BEISPIEL 6 (6-2).
-
[SEQ ID NR. 32]
-
Diese
zeigt die Basensequenz des Primers, verwendet in dem später beschriebenen
BEISPIEL 7.
-
[SEQ ID NR. 33]
-
Diese
zeigt die Basensequenz des Primers, verwendet in dem später beschriebenen
BEISPIEL 7.
-
[SEQ ID NR. 34]
-
Diese
zeigt die Aminosäuresequenz
vom Schlangengift MIT1, gereinigt in dem später beschriebenen BEISPIEL
8.
-
[SEQ ID NR. 35]
-
Diese
zeigt die Basensequenz der cDNA, die das menschentypische I5E-Rezeptorprotein
kodiert.
-
[SEQ ID NR. 36]
-
Diese
zeigt die Aminosäuresequenz
des menschentypischen I5E-Rezeptorproteins.
-
[SEQ ID NR. 37]
-
Diese
zeigt die Basensequenz des Primers, verwendet in dem später beschriebenen
BEISPIEL 9.
-
[SEQ ID NR. 38]
-
Diese
zeigt die Basensequenz des Primers, verwendet in dem später beschriebenen
BEISPIEL 9.
-
[SEQ ID NR. 39]
-
Diese
zeigt die Basensequenz der cDNA, die das neue G-Protein-gekoppelte
Rezeptorprotein (rZAQ1) kodiert.
-
[SEQ ID NR. 40]
-
Diese
zeigt die Aminosäuresequenz
des neuen G-Protein-gekoppelten Rezeptorproteins (rZAQ1).
-
[SEQ ID NR. 41]
-
Diese
zeigt die Basensequenz des Primers, verwendet in dem später beschriebenen
BEISPIEL 10.
-
[SEQ ID NR. 42]
-
Diese
zeigt die Basensequenz des Primers, verwendet in dem später beschriebenen
BEISPIEL 10.
-
[SEQ ID NR. 43]
-
Diese
zeigt die Basensequenz des Primers, verwendet in dem später beschriebenen
BEISPIEL 10.
-
[SEQ ID NR. 44]
-
Diese
zeigt die Basensequenz des Primers, verwendet in dem später beschriebenen
BEISPIEL 10.
-
[SEQ ID NR. 45]
-
Diese
zeigt die Basensequenz des Primers, verwendet in dem später beschriebenen
BEISPIEL 10.
-
[SEQ ID NR. 46]
-
Diese
zeigt die Basensequenz der cDNA, die das neue G-Protein-gekoppelte
Rezeptorprotein (rZAQ2) kodiert.
-
[SEQ ID NR. 47]
-
Diese
zeigt die Aminosäuresequenz
des neuen G-Protein-gekoppelten Rezeptorproteins (rZAQ2)
-
[SEQ ID NR. 48]
-
Diese
zeigt die Aminosäuresequenz
des Maus-abgeleiteten G-Protein-gekoppelten Rezeptorproteins (GPR73).
-
[SEQ ID NR. 49]
-
Diese
zeigt die Aminosäuresequenz
des Maus-abgeleiteten G-Protein-gekoppelten Rezeptorproteins (mI5E).
-
[SEQ ID NR. 50]
-
Diese
zeigt die Basensequenz der cDNA, die das Maus-abgeleitete G-Protein-gekoppelte
Rezeptorprotein (GPR73) kodiert.
-
[SEQ ID NR. 51]
-
Diese
zeigt die Basensequenz der cDNA, die das Maus-abgeleitete G-Protein-gekoppelte
Rezeptorprotein (mI5E) kodiert.
-
[SEQ ID NR. 52]
-
Diese
zeigt die Basensequenz des DNA-Fragmentes #1, verwendet in REFERENZBEISPIEL
1.
-
[SEQ ID NR. 53]
-
Diese
zeigt die Basensequenz des DNA-Fragmentes #2, verwendet in REFERENZBEISPIEL
1.
-
[SEQ ID NR. 54]
-
Diese
zeigt die Basensequenz des DNA-Fragmentes #3, verwendet. in REFERENZBEISPIEL
1.
-
[SEQ ID NR. 55]
-
Diese
zeigt die Basensequenz des DNA-Fragmentes #4, verwendet in REFERENZBEISPIEL
1.
-
[SEQ ID NR. 56]
-
Diese
zeigt die Basensequenz des DNA-Fragmentes #5, verwendet in REFERENZBEISPIEL
1.
-
[SEQ ID NR. 57]
-
Diese
zeigt die Basensequenz des DNA-Fragmentes #6, verwendet in REFERENZBEISPIEL
1.
-
[SEQ ID NR. 58]
-
Diese
zeigt die Basensequenz der synthetischen DNA, die den menschentypischen
ZAQ-Liganden kodiert,
der durch SEQ ID NR. 21 dargestellt ist.
-
Die
Transformante Escherichia coli DH5α/pCR2.1-ZAQC, erhalten in dem
später
beschriebenen BEISPIEL 1, ist im National Institute of Advanced
Industrial Science and Technology, International Patent Organism
Depositary (jetzt überholt
Ministry of International Trade and Industry, Agency of Industrial
Science and Technology, National Institute of Bioscience and Human
Technology (NIBH)), mit Sitz im Central 6, 1-1-1 Higashi, Tsukuba,
Ibaraki, Japan, unter der Zugangsnummer FERM BP-6855 seit 23. August
1999 und im Institute for Fermentation, Osaka (IFO), mit Sitz in
2-17-85, Juso-honmachi, Yodogawa-ku, Osaka-shi, Osaka, Japan, unter
der Zugangsnummer IFO 16301 seit 4. August 1999 hinterlegt.
-
Die
Transformante Escherichia coli DH5α/pCR2.1-ZAQT, erhalten in dem
später
beschriebenen BEISPIEL 1, ist im National Institute of Advanced
Industrial Science and Technology, International Patent Organism
Depositary (jetzt überholt
Ministry of International Trade and Industry, Agency of Industrial
Science and Technology, National Institute of Bioscience und Human
Technology (NIBH)) unter der Zugangsnummer FERM BP-6856 seit 23.
August 1999 und im Institute for Fermentation, Osaka (IFO) unter
der Zugangsnummer IFO 16302 seit 4. August 1999 hinterlegt.
-
Die
Transformante Escherichia coli TOP10/pHMITA, erhalten in dem später beschriebenen
BEISPIEL 4, ist im National Institute of Advanced Industrial Science
and Technology, International Patent Organism Depositary (jetzt überholt
Ministry of International Trade and Industry, Agency of Industrial
Science and Technology, National Institute of Bioscience and Human
Technology (NIBH)) unter der Zugangsnummer FERM BP-7219 seit 13.
Juli 2000 und im Institute for Fermentation, Osaka (IFO) unter der
Zugangsnummer IFO 16440 seit 26. Mai 2000 hinterlegt.
-
Die
Transformante Escherichia coli TOP10/pHMITG, erhalten in dem später beschriebenen
BEISPIEL 4, ist im National Institute of Advanced Industrial Science
and Technology, International Patent Organism Depositary (jetzt überholt
Ministry of International Trade and Industry, Agency of Industrial
Science and Technology, National Institute of Bioscience and Human
Technology (NIBH)) unter der Zugangsnummer FERM BP-7220 seit 13.
Juli 2000 und im Institute for Fermentation, Osaka (IFO) unter der
Zugangsnummer IFO 16441 seit 26. Mai 2000 hinterlegt.
-
Die
Transformante Escherichia coli DH5α/pCR2.1-rZAQ1, erhalten in dem
später
beschriebenen BEISPIEL 9, ist im National Institute of Advanced
Industrial Science und Technology, International Patent Organism
Depositary (jetzt überholt
Ministry of International Trade and Industry, Agency of Industrial
Science and Technology, National Institute of Bioscience and Human
Technology (NIBH)) unter der Zugangsnummer FERM BP-7275 seit 21.
August 2000 und im Institute for Fermentation, Osaka (IFO) unter
der Zugangsnummer IFO 16459 seit 1. August 2000 hinterlegt.
-
Die
Transformante Escherichia coli DH10B/pCMV-rZAQ2, erhalten in dem
später
beschriebenen BEISPIEL 10, ist im National Institute of Advanced
Industrial Science und Technology, International Patent Organism
Depositary (jetzt überholt
Ministry of International Trade and Industry, Agency of Industrial
Science and Technology, National Institute of Bioscience und Human
Technology (NIBH)) unter der Zugangsnummer FERM BP-7276 seit 21.
August 2000 und im Institute for Fermentation, Osaka (IFO) unter
der Zugangsnummer IFO 16460 seit 1. August 2000 hinterlegt.
-
Die
Transformante Escherichia coli MM294 (DE3)/pTCh1ZAQ, erhalten in
dem später
beschriebenen REFERENZBEISPIEL 1, ist im National Institute of Advanced
Industrial Science und Technology, International Patent Organism
Depositary unter der Zugangsnummer FERM BP-7571 seit 27. April 2001
und im Institute for Fermentation, Osaka (IFO) unter der Zugangsnummer
IFO 16527 seit 16. Januar 2001 hinterlegt.
-
BEISPIELE
-
Hierin
nachstehend wird die vorliegende Erfindung ausführlich in bezug auf die BEISPIELE
und REFERENZBEISPIELE beschrieben, aber soll den Umfang der vorliegenden
Erfindung nicht darauf beschränken. Die
Genmanipulationsverfahren unter Verwendung von Escherichia coli
wurden gemäß den Verfahren
durchgeführt,
die in der Molekularen Klonierung beschrieben sind.
-
BEISPIEL 1
-
Klonen der cDNA die das
G-Protein-gekoppelte Rezeptorprotein ZAQ kodiert, und Bestimmung
der Basensequenz
-
Unter
Verwendung der menschlichen hypophysären cDNA (CLONTECH Laboratories,
Inc.) als eine Matrize und zwei Primern, nämlich Primer 1
(5'-GTC GAC ATG GAG
ACC ACC ATG GGG TTC ATG G-3';
SEQ ID NR:4)
und Primer 2
(5'-ACT AGT TTA TTT TAG TCT GAT GCA GTC
CAC CTC TTC-3';
SEQ ID NR:5) wurde PCR durchgeführt. Die
Reaktionslösung
in der obigen Reaktion bestand aus 1/10 Volumen der oben beschriebenen
cDNA, 1/50 Volumen von Advantage 2 Polymerase Mix (CLONTECH Laboratories,
Inc.), 0,2 μM
Primer 1, 0,2 μM
Primer 2, 200 μM
dNTPs und einem Puffer, zugeführt
mit dem Enzym, um das Endvolumen von 25 μl herzustellen. Bei der PCR
wurde, nachdem die Reaktionslösung
bei 94 °C
für 2 Minuten
erhitzt wurde, eine Zyklusgruppe bei 94 °C für 20 Sekunden, gefolgt von
72 °C für 100 Sekunden
dreimal wiederholt, wurde eine Zyklusgruppe bei 94 °C für 20 Sekunden
und bei 68 °C
für 100
Sekunden dreimal wiederholt, wurde eine Zyklusgruppe bei 94 °C für 20 Sekunden,
bei 64 °C
für 20
Sekunden und bei 68 °C
für 100
Sekunden 38 Mal wiederholt, und schließlich wurde eine Extensionsreaktion bei
68 °C für 7 Minuten
durchgeführt.
Nach Beendigung der PCR wurde das Reaktionsprodukt zu dem Plasmidvektor
pCR2.1 (Invitrogen, Inc.) gemäß den Instruktionen,
die dem TA-Klonierungskit
beigefügt
sind (Invitrogen, Inc.), subkloniert. Dann wurde es in Escherichia
coli DH5α eingeführt, und
die Klone, die die cDNA trugen, wurden in LB-Agarmedium, das Ampicillin
enthält,
selektiert. Die Sequenz von jedem Klon wurde analysiert, wodurch
zwei cDNA-Sequenzen erhalten wurden, die das neue G-Protein-gekoppelte
Rezeptorprotein kodierten, d. h., ZAQC (SEQ ID NR. 2) und ZAQT (SEQ
ID NR. 3). Die Proteine mit der Aminosäuresequenz, die aus dieser
cDNA abgeleitet wurde, wurde als ZAQ bezeichnet, da sie alle dieselbe
Basensequenz haben (SEQ ID NR. 1). Die Transformante, die die DNA
trägt,
die durch SEQ ID NR. 2 dargestellt ist, wurde Escherichia coli DH5α/pCR2.1-ZAQC
genannt, und die Transformante, die die DNA trägt, die durch SEQ ID NR. 3
dargestellt ist, wurde Escherichia coli DH5α/pCR2.1-ZAQT genannt.
-
BEISPIEL 2
-
Analyse der
Expressionsverteilun von ZAQ durch Taqman PCR
-
Die
Primer und eine Sonde, die in Taqman PCR verwendet werden sollte,
wurden unter Verwendung von Primer Express Ver. 1.0 (PE Biosystems
Japan) untersucht, und Primer 3
(5'-TCATGTTGCTCCACTGGAAGG-3' (SEQ ID (NR: 6)),
Primer
4
(5'-CCAATTGTCTTGAGGTCCAGG-3' (SEQ ID NR: 7))
und
ZAQ-Sonde
(5'-TTCTTACAATGGCGGTAAGTCCAGTGCAG-3' (SEQ ID NR: 8))
wurden selektiert. FAM (6-Carboxyfluorescein) wurde als ein Reporterfarbstoff
für die
Sonde zugegeben.
-
Das
PCR-Fragment, das unter Verwendung von pAK-ZAQC als eine Matrize,
und Primer ZAQC Sal (5'-GTCGACATGGAGACCACCATGGGGTTCATGG-3'(SEQ ID NR: 9)) und
Primer ZAQC Spe (5'-ACTAGTTTATTTTAGTCTGATGCAGTCCACCTCTTC-3' (SEQ ID NR:10))
amplifiziert wurde, wurde mit CHROMA SPIN200 gereinigt (CLONTECH
Laboratories, Inc. (CA,USA)), und dann auf eine Konzentration von
100–106 Kopien/μl
zur Verwendung als eine Standard-DNA eingestellt. Menschliche Multiple
Tissue cDNA Panel I und Panel II (CLONTECH Laboratories, Inc.) wurden
als die cDNA-Quelle für
jedes Gewebe verwendet. Zu den Primern, der Sonde und Matrize wurde
Taqman Universal PCR Master Mix (PE Bio systems Japan) in der Menge
zugegeben, die durch die beiliegenden Instruktionen angegeben werden,
und dann wurden PCR und Analyse mit ABI PRISM 7700 Sequenz Detection
System (PE Biosystems Japan) durchgeführt.
-
Die
Ergebnisse werden in 8 und Tabelle 1 gezeigt. Die
Expression von ZAQ wurde hauptsächlich in
den Hoden gefunden, und als nächstes
an den Stellen, wie Lunge, Gehirn und andere.
-
-
BEISPIEL 3
-
Isolation von ZAQ-aktivierendem
Peptid
-
(3-1) Herstellung von
Milchextraktlösung
-
Unter
Verwendung von kommerziell erhältlicher
Milch, die bei einer niedrigen Temperatur pasteurisiert wurde, wurden
die folgenden. Verfahrensweisen durchgeführt, um eine Extrakt lösung herzustellen.
Zwei Liter Milch wurden bei 10.000 U/min für 15 Minuten bei 4 °C mit einer
Hochgeschwindigkeitszentrifuge zentrifugiert (CR26H, R1OA Rotor:
Hitachi, Ltd.). Der erhaltene Überstand
wurde durch Gaze filtriert, wodurch Lipide entfernt wurden. Essigsäure wurde
zu dem Überstand
zugegeben, um die Konzentration auf die Endkonzentration von 1 M
einzustellen, und das Gemisch wurde für 30 Minuten bei 4 °C gerührt. Dann
wurde das Gemisch bei 10.000 U/min für 15 Minuten mit einer Hochgeschwindigkeitszentrifuge
zentrifugiert (CR26H R10A Rotor: Hitachi System Engineering Co.,
Ltd.). Der erhaltene Uberstand wurde filtriert, wodurch unlösliche Stoffe
entfernt wurden. Während
des Rührens
wurde Aceton doppelt so viel wie das Volumen des Überstandes
dazugegeben. Das Rühren
wurde bei 4 °C
für 3 Stunden
fortgesetzt. Dann wurde das Gemisch bei 10.000 U/min für 15 Minuten
mit einer Hochgeschwindigkeitszentrifuge zentrifugiert (CR26H R10A
Rotor: Hitachi, Ltd.). Der erhaltene Überstand wurde filtriert, wodurch
die unlöslichen
Stoffe entfernt wurden. Der erhaltene Überstand wurde einem Rotationsverdampfer
zugeführt,
wodurch Aceton entfernt wurde, und auf 1350 ml Endvolumen konzentriert.
Dann wurden jeweils 675 ml des resultierenden Konzentrats mit 338
ml Diethylether gemischt, und das Gemisch wurde in einem Trenntrichter
kräftig
geschüttelt,
wodurch es in zwei Phasen getrennt wurde, um die wässerige
Phase zu sammeln. Dieselbe Verfahrensweise wurde einmal in bezug
auf die erhaltene wässerige Phase
wiederholt, wodurch eine klare wässerige
Lösung
erhalten wurde. Die erhaltene wässerige
Lösung
wurde auf 800 ml unter Verwendung eines Rotationsverdampfers konzentriert,
wodurch ein Endextrakt erhalten wurde.
-
(3-2) Grobfraktionierung
der Milchextraktlösung
unter Verwendung von C18-Umkehrphasenchromatographie
-
Methanol
wurde zu 10 g einer Sep-Pak C18-Säule (Waters), gepackt mit Octadecylgruppefixiertem Kieselgel,
zugegeben, wodurch das Gel quoll. Die Säule wurde dann mit 1 M Essigsäure äquilibriert.
Die Extraktlösung,
hergestellt in (3-1) (die Extraktlösung aus 2 Liter Milch), wurde
auf die Säule
geladen. Dann wurden 100 ml 1 M Essigsäure durch die Säule geführt, wodurch
das Gel gewaschen wurde. Dann wurde die Säule mit 200 ml von 60 % Acetonitril/0,1
% Trifluoressigsäure
eluiert, wodurch die gewünschte
rohe Peptidkomponente eluiert wurde. Nachdem das erhaltene Eluat
unter Verwendung eines Rotationsverdampfers konzentriert wurde,
wurde das Konzentrat mit einem Gefriertrockner lyophilisiert (12EL;
VirTis).
-
(3-3) Grobfraktionierung
der Milchextraktlösung
durch Sulfopropyl-Ionenaustauschchromatographie
-
SP
Sephadex C-25 (Amersham Pharmacia Biotech), das in 100 mM HCl quoll,
wurde auf eine Säule aus
Polypropylen in einem Volumen von 2 ml geladen. Die Säule wurde
mit destilliertem Wasser und Ammoniumformiat (pH 4,0) gewaschen
und mit Elutionsmittel I äquilibriert
(2 M Ammoniumformiat : Acetonitril : Wasser = 1 : 25 : 74). Das
lyophilisierte Produkt, das in (3-2) erhalten wurde, wurde in 20
ml Elutionsmittel I gelöst. Die
resultierende Lösung
wurde auf 2 ml SP Sephadex C-25 geladen. Nachdem die Säule mit
10 ml Elutionsmittel I gewaschen wurde, wurde das Peptid mit jeweils
10 ml Elutionsmittel II (2 M Ammoniumformiat : Acetonitril : Wasser
= 1 : 2,5 : 6,5), Elutionsnittel III (2 M Ammoniumformiat : Acetonitril
: Wasser = 1 : 1 : 2) und Elutionsmittel IV (2 M Ammoniumformiat
: Acetonitril : Wasser = 1 : 0,5 : 0,5) in dieser Reihenfolge gewaschen. Jedes
der Eluate I bis IV wurde mit einem Gefriertrockner lyophilisiert
(12EL; VirTis).
-
(3-4) Fraktionierung des
Milchextraktes durch TSKgel-ODS80Ts-Umkehrphasen-Hochleistungsflüssigchromatographie
-
Mittels
Führen
von 91,7 Vol.-% Elutionsmittel A (0,1 % Trifluoressigsäure/destilliertes
Wasser) und 8,3 Vol.-% Elutionsmittel B (0,1 % Trifluoressigsäure/60 %
Acetonitril) durch eine Säule
für TSKgel-ODS80Ts-Umkehrphasen-Hochleistungsflüssigchromatographie
(Toso Co., Ltd., 4,6 mm × 25
cm) bei einer Fließgeschwindigkeit
von 1 ml/min bei 40 °C
wurde die Säule äquilibriert.
Die lyophilisierten Produkte der Eluate I bis IV, erhalten in (3-3),
wurden in 4 ml 1 M Essigsäure
gelöst,
und dann der Chromatographiebehandlung unterzogen. Das heißt, 4 ml
der Lösung
des lyophilisierten Produktes wurden auf die Säule geladen, und die Konzentration des
Elutionsmittels B wurde auf 67 Vol.-% Elutionsmittel A/33 Vol.-%
Elutionsmittel B über
1 Minute erhöht
und dann mit einem linearen Gradienten von 67 Vol.-% Elutionsmittel
A/33 Vol.-% Elutionsmittel B auf 0 Vol.-% Elutionsmittel A/100 Vol.-%
Elutionsmittel B über
die nächsten
40 Minuten bei einer Fließgeschwindigkeit
von 1 ml/min erhöht.
-
Jeweils
1 ml Eluat wurde entnommen, wobei jeder Fraktion eine Fraktionsnummer
verliehen wurde, und 2 μl
von jeder Fraktion wurden mit 150 μl von 0,2 % Rinderserumalbumin
(BSA)/destilliertem Wasser gemischt, gefolgt von Lyophilisierung.
Das lyophilisierte Produkt wurde als eine Assayprobe zum Messen
der die intrazelluläre
Ca2+-Ionenkonzentration erhöhenden Aktivität verwendet,
die später
in (3-5) beschrieben wurde.
-
(3-5) Messung der die
intrazelluläre
Ca2+-Ionenkonzentration erhöhenden Aktivität unter
Verwendung von FLIPR
-
Die
Zellinie, die ZAQ stabil exprimieren kann, wurde folgendermaßen hergestellt.
Das heißt,
ein Klon von DH5α/pCR2.1-ZAQC,
erhalten in BEISPIEL 1, wurde in LB-Medium, das mit Ampicillin ergänzt wurde, während kräftigen Schüttelns inkubiert,
wodurch Plasmid pCR2.1-ZAQC
erhalten wurde. Das Plasmid wurde mit Restriktionsenzymen Sal I
und Spe I digeriert, um den Insert-Teil, der ZAQC kodiert, zu exzidieren.
Dann wurde pAKKO-1.11H (Biochemica et Biophysica Acta, 1219 (1994)
251–259),
das ebenso mit Restriktionsenzymen Sal I und Spe I digeriert wurde,
mit dem Insert-Teil unter Verwendung von Ligation Express Kit ligiert (CLONTECH
Laboratories, Inc. (CA, USA)), was zu E. coli DH10B durch das Elektroporationsverfahren
transfektiert wurde. Die Struktur des Plasmids, das in dem erhaltenen
Klon enthalten ist, wurde durch Restriktionsenzymbehandlung und
Sequenzanalyse analysiert. Das korrekt konstruierte Plasmid wurde
als Plasmid pAK-ZAQC zur CHO-Zellexpression verwendet.
-
Dieses
Plasmid pAK-ZAQC wurde zu einer CHO/dhfr–-Zelle
(American Type Culture Collection) unter Verwendung von CellPhect
Transfection Kit (Amersham Pharmacia Biotech) transfektiert. Zunächst wurden 120 μl Puffer
A (CellPhect Transfection Kit) zu einer Lösung aus 4 μg Plasmid-DNA in 120 μl destilliertem
Wasser zugegeben, und das Gemisch wurde gerührt. Nach dem Stehenlassen
für 10
Minuten wurden 240 μl
Puffer B (CellPhect Transfection Kit) zu dem Gemisch zugegeben.
Das Gemisch wurde kräftig
gerührt,
wodurch der DNA-Calciumphosphatkomplex gebildet wurde, der die DNA
enthielt. 5 × 105 CHO/dhfr–-Zellen wurden auf
einer 60-mm-Petrischale inokuliert. Nach der Inkubation in Ham-F-12-Medium (Nissui Pharmaceutical
Co., Ltd.), ergänzt
mit 10 % fetalem Rinderserum (BIO WHITTAKER, Inc.), wurden bei 37 °C unter 5
% CO2 für
einen Tag 480 μl
einer Suspension aus dem DNA-Calciumphosphatkomplex tropfenweise
zu den Zellen auf der Petrischale zugegeben. Nach der Inkubation
bei 37 °C
unter 5 % CO2 für 6 Stunden wurden die Zellen
zweimal mit serumfreien Ham-F-12-Medium gewaschen, und 1,2 ml Puffer
(140 mM NaCl, 25 mM HEPES, 1,4 mM Na2HPO4, pH 7,1), ergänzt mit 15 % Glycerol, wurden
zu den Zellen auf der Schale zugegeben, um die Zellen für 2 Minuten
zu behandeln. Die Zellen wurden erneut zweimal mit serumfreien Ham-F-12-Medium
gewaschen, gefolgt von der Inkubation in Ham-F-12-Medium, enthaltend
10 % fetales Rinderserum, bei 37 °C
unter 5 % CO2 über Nacht. Die Zellen wurden
mit Trypsin zur Dispersion behandelt, aus der Schale rückgewonnen, und
jeweils 2 × 104 wurden in einer 6-Lochplatte inokuliert.
Die Inkubation wurde in Dulbecco's
Modifiziertem Eagle-Medium (DMEM), ergänzt mit 10 % dialysiertem fetalem
Rinderserum (JRH BIOSCIENCES), 1 mM MEM nicht-essentieller Aminosäurelösung (Dainippon
Pharmaceutical Co., Ltd.), 100 Einheiten/ml Penicillin und 100 μg/ml Streptomyzin,
bei 37 °C
unter 5 % CO2 initiiert. Die transformierten
CHO-Zellen, in die das Plasmid eingeführt wurde, konnten in dem Medium überleben,
aber nicht-transfektierte Zellen starben allmählich ab. Daher wurde das Medium
ausgetauscht, wodurch die toten Zellen alle 2 Tage nach der Initiierung
der Inkubation entfernt wurden. Etwa 21 Kolonien. der transformierten
CHO-Zellen, die nach 8 bis 10 Tagen der Inkubation wuchsen, wurden
ausgewählt.
RNA wurde aus jeder der ausgewählten
Zellen unter Verwendung eines kommerziell erhältlichen RNA-Isolationskits
gewonnen, gefolgt von dem öffentlich
bekannten RT-PCR-Verfahren, um den ZAQ-Expressions-CHO-Zellen-B-1-Klon
auszuwählen
(hierin nachstehend nur als ZAQC-B1-Zelle bezeichnet), der hohe
Expression von ZAQ zeigt.
-
Zur
Kontrolle wurde ETA-(Endothelin-A-Rezeptor)-exprimierender CHO-Zellen-Klon
Nr. 24 (hierin nachstehend nur als ETA24-Zelle bezeichnet; siehe
Journal of Pharmacology and Experimental Therapeutics, 279, 675–685, 1996)
verwendet.
-
Die
die intrazelluläre
Ca2+-Ionenkonzentration erhöhende Aktivität von ZAQC-B1-Zellen
oder ETA24-Zellen wurde für
die in (3-4) oben erhaltenen Proben unter Verwendung des FLIPR analysiert
(Molecular Devices, Inc.). Die ZAQC-B1-Zellen und ETA24-Zellen,
die beide in DMEM-Medium, ergänzt
mit 10 % dialysiertem fetalem Rinderserum (hierin nachstehend als
dFBS bezeichnet), subkultiviert wurden, wurden verwendet. Die ZAQC-B1-Zellen und ETA24-Zellen
wurden jeweils in dem Medium (10 % dFBS-DMEM) suspendiert, um die
Konzentration auf 15 × 104 Zellen/ml einzustellen. Ein aliquoter Teil
von 200 μl
von jeder Zellsuspension wurde auf jedem Loch (3,0 × 104 Zellen/200 μl/Loch) in einer 96-Lochplatte
für FLIPR
inokuliert (Black Plate Clear Bottom, Coster), die in einem Inkubator
bei 37 °C
unter 5 % CO2 über Nacht inkubiert wurde,
und die so erhaltenen Zellen wurden verwendet (hierin nachstehend
als die Zellplatte bezeichnet). Zwanzig Milliliter von H/HBS (9,8
g Nissui Hanks 2 (Nissui Pharmaceutical Co., Ltd.), 0,35 g Natriumhydrogencarbonat, 4,77
g HEPES, eingestellt auf pH 7,4 mit einer Natriumhydroxidlösung, gefolgt
von Sterilisation durch einen Sterilisationsfilter), 200 μl von 250
mM Probenecid und 200 μl
fetales Rinderserum (FBS) wurden gemischt. Ebenso wurden 2 Phiolen
(50 μg)
Fluo 3-AM (Dojin Chemical Research Institute) in 40 μl Dimethylsulfoxid
und 40 μl von
20%iger Pluronsäure
(Molecular Probes, Inc.) gelöst,
und die resultierende Lösung
wurde zu dem obigen H/HBSS-Probenecid-FBS-Gemisch
zugegeben. Nach dem Mischen wurde das Medium von der Zellplatte
entfernt, und jeweils 100 μl
des Gemisches wurden in jedem Loch der Zellplatte unter Verwendung
einer 8-Kanal-Pipette dispensiert. Die Zellplatte wurde dann bei
37 °C unter
5 % CO2 für eine Stunde (Farbstoffladung) inkubiert.
Für die
Assayproben, erhalten in dem oben beschriebenen BEISPIEL (3-4),
wurden 150 μl
H/HBSS, enthaltend 2,5 mM Probenecid und 0,1 % CHAPS, zu jeder Fraktion
zur Verdünnung
zugegeben. Die verdünnte
Lösung
wurde auf eine 96-Lochplatte für
FLIPR übertragen
(V-Bottom Plate, Coster, hierin nachstehend als die Probenplatte
bezeichnet). Nach Beendigung der Farbstoffladung auf die Zellplatte
wurde die Zellplatte viermal mit dem Waschpuffer, bestehend aus
H/HBSS, ergänzt
mit 2,5 mM Probenedid, unter Verwendung eines Plattenwäschers gewaschen
(Molecular Devices, Inc.). Nach dem Waschen wurden 100 μl des Waschpuffers für weitere
Verfahren sichergestellt. Diese Zellplatte und die Probenplatte
wurden auf FLIPR gegeben, um einen Assay durchzuführen (durch
FLIPR wurden 50 μl
der Probe aus der Probenplatte zu der Zellplatte übertragen).
-
Infolgedessen
wurde die die intrazelluläre
Ca2+-Ionenkonzentration erhöhende Aktivität, die für ZAQC-B1-Zellen
spezifisch ist, in Fraktion Nr. 53 gefunden, die durch Anwenden
von oben beschriebenen Elutionsmittel IV (3-3) auf die Umkehrphasen-Hochleistungsflüssigchromatographie
in (3-4) oben fraktioniert wurde.
-
(3-6) Reinigung unter
Verwendung von TSKgel-Super-Phenyl-Umkehrphasen-Hochleistungsflüssigchromatographie
-
(1)
Mittels Führen
von 91,7 Vol.-% Elutionsmittel A (0,1 % Trifluoressigsäure/destilliertes
Wasser)/8,3 Vol.-% Elutionsmittel B (0,1 % Trifluoressigsäure/60 %
Acetonitril) durch eine Säule
zur TSKgel-Super-Phenyl-Umkehrphasen-Hochleistungsflüssigchromatographie
(Toso Co., Ltd., 0,46 cm × 10
cm) bei einer Fließgeschwindigkeit
von 1 ml/min bei 40 °C
wurde die Säule äquilibriert.
Die Fraktion Nr. 53, erhalten in oben beschriebenen (3-4), wurde
der Chromatographie unterzogen. Das heißt, 1 ml der Fraktion Nr. 53
wurde auf die Säule
gela den, und die Konzentration an Elutionsmittel B wurde auf 75
Vol.-% Elutionsmittel A/25 Vol.-% Elutionsmittel B über 1 Minute
erhöht
und dann mit einem linearen Gradienten auf 67 Vol.-% Elutionsmittel
A/33 Vol.-% Elutionsmittel B über
75 Minuten bei einer Fließgeschwindigkeit
von 1 ml/min erhöht.
-
Jeweils
500 μl Eluat
wurde entnommen, wobei jeder Fraktion eine Fraktionsnummer verliehen
wurde, und 25 μl
jeder fraktionierten Fraktion wurden mit 150 μl 0,2 % BSA gemischt, gefolgt
durch Lyophilisierung mit einem Gefriertrockner (12EL; VirTis).
Das lyophilisierte Produkt wurde mit 150 μl H/HBSS, enthaltend 2,5 mM Probenecid
und 0,1 % CHAPS, zugegeben, wodurch das lyophilisierte Produkt gelöst wurde.
Unter Verwendung von 50 μl
der resultierenden Lösung
wurde die die intrazelluläre
Ca2+-Ionenkonzentration erhöhende Aktivität durch
das in (3-5) oben beschriebene Verfahren gemessen, wodurch die Wirkung
der Aktivierung des Rezeptors zu den ZAQC-B1-Zellen analysiert wurde.
Das Ergebnis offenbart, daß die
Komponente mit der Wirkung zum Aktivieren des Rezeptors für die Ziel-ZAQC-B1-Zellen, d.
h., die ZAQ-aktivierende Komponente, hauptsächlich in Fraktion Nr. 103–105 eluiert
wurde.
-
(3-7) Reinigung unter
Verwendung von μRPC
C2/C18 ST4.6/100 Umkehrphasen-Hochleistungsflüssigchromatographie
-
Mittels
Führen
von 95 Vol.-% Elutionsmittel A (Heptafluorbuttersäure/destilliertes
Wasser)/5 Vol.-% Elutionsmittel B (0,1 % Heptafluorbuttersäure/100
% Acetonitril) durch eine Säule
zur μRPC
C2/C18 ST4.6/100 Umkehrphasen-Hochleistungsflüssigchromatographie (Amersham
Pharmacia Biotech, 0,46 cm × 10
cm) bei einer Fließgeschwindigkeit
von 1 ml/min bei 40 °C
wurde die Säule äquilibriert.
-
Nachdem
die Fraktionen Nr. 103–105,
ausgewählt
aus den Fraktionen, die in (3-6) oben durch TSKgel-Super-Phenyl-Umkehrphasen-Hochleistungsflüssigchromatographie
erhalten wurden, direkt auf die μRPC
C2/C18 ST4.6/100-Umkehrphasensäule
geladen wurden, wurde die Elution durch schnelles Erhöhen von
95 Vol.-% Elutionsmittel A (0,1 % Heptafluorbuttersäure/destilliertes
Wasser)/5 Vol.-% Elutionsmittel B (0,1 % Heptafluorbuttersäure/100
% Acetonitril) auf 65 Vol.-% Elutionsmittel A/35 Vol.-% Elutionsmittel
B bei einer Fließgeschwindigkeit
von 1 ml/min über
1 Minute und dann durch Erhöhen
mit einem linearen Gradienten auf 50 Vol.-% Elutionsmittel A/50
Vol.-% Elutionsmittel B über
die nächsten
60 Mi nuten durchgeführt,
wodurch das Eluat gewonnen wurde. Das Eluat wurde als ein einzelner
Peak bei 210 nm durch UV-Absorption detektiert.
-
Jeweils
500 μl Eluat
wurden entnommen, wobei jeder Fraktion eine Fraktionsnummer verliehen
wurde, und jeweils 25 μl
aus den erhaltenen Fraktionen wurden mit 150 μl 0,2 % BSA gemischt, gefolgt
von Lyophilisierung mit einem Gefriertrockner (12EL; VirTis). Das
lyophilisierte Produkt wurde mit 150 μl H/HBSS, enthaltend 2,5 mM
Probenecid und 0,1 % CHAPS, zugegeben, wodurch das lyophilisierte
Produkt gelöst
wurde. Unter Verwendung von 50 μl
der Lösung
wurde die Rezeptoraktivierungsaktivität auf die ZAQ-C-B1-Zellen durch das
in (3-5) oben beschriebene Testverfahren analysiert. Das Ergebnis
offenbarte, daß die
Komponente mit der Rezeptoraktivierungsaktivität auf die jeweiligen ZAQ-C-B1-Zellen,
nämlich
die ZAQ-aktivierende Komponente, hauptsächlich in Fraktionen Nr. 82
bis 84 eluiert wurde. Dieser Aktivierungspeak stimmte vollständig mit dem
UV-Absorptionspeak bei 210 nm überein,
was zu der Schlußfolgerung
führt,
daß das
Produkt genug gereinigt wurde, um das einzelne Peptid zu erhalten.
-
(3-8) Analyse der Struktur
von gereinigtem ZAQ-aktivierendem Peptid
-
Das
folgende Verfahren wurde verwendet, um die Struktur der ZAQ-aktivierenden
Komponente zu bestimmen, die in dem oben beschriebenen BEISPIEL
(3-7) erhalten wurde. Die gereinigte ZAQ-aktivierende Komponente
wurde lyophilisiert, und das lyophilisierte Produkt wurde in Lösungsmittel
DMSO (Dimethylsulfoxid) gelöst.
Ein aliquoter Teil der Lösung
wurde für
das N-terminale Aminosäuresequenzieren
unter Verwendung eines Proteinsequenzers bereitgestellt (Perkin-Elmer,
Inc., PE Biosystems Procise 491cLC). Infolgedessen konnten 14 Reste
von den Aminosäureresten
von dem N-Terminus bis zu dem 16. Aminosäurerest identifiziert werden
(Ala Val Ile Thr Gly Ala Xaa Glu Arg Asp Val Gln Xaa Arg Ala Gly
(SEQ ID NR. 11; Xaa ist ein nicht-identifizierter Rest)).
-
BEISPIEL 4
-
Klonen der
cDNA für
menschentypisches ZAQ-Ligandenpeptid
-
Eine
Blast-Suche wurde in einer Datenbank unter Verwendung der N-terminalen
Aminosäuresequenz (SEQ
ID NR. 11) des gereinigten ZAQ-aktivierenden Peptids, das aus Milch
in BEISPIEL 3 extrahiert wurde, als Abfrage durchgeführt. So
wurde menschliches EST (X40467) entdeckt, das dieselbe Basensequenz
wie die der DNA aufweist, die das Peptid mit der Aminosäuresequenz
kodiert, die durch. SEQ ID NR. 11 dargestellt ist. Da diese Sequenz
kein vollständiges
offenes Leseraster aufweist, wurde die nicht-identifizierte Sequenz durch
das RACE-Verfahren identifiziert, und folglich wurde der cDNA-Klon
mit dem vollständigen
offenen Leseraster erhalten.
-
Basierend
auf den Informationen aus EST (X40467) wurden Primer ZF1 (SEQ ID
NR. 12), ZF2 (SEQ ID NR. 13) und ZF3 (SEQ ID NR. 14) konstruiert,
und das 3'-RACE-Experiment
wurde unter Verwendung von menschlicher Testis-Marathon-Ready-cDNA
(CLONTECH Laboratories, Inc.) als eine Matrize durchgeführt.
ZF1:
5'-GGTGCCACGCGAGTCTCAATCATGCTCC-3' (SEQ ID Nr.: 12)
ZF2:
5'-GGGGCCTGTGAGCGGGATGTCCAGTGTG-3' (SEQ ID Nr.: 13)
ZF3:
5'-CTTCTTCAGGAAACGCAAGCACCACACC-3' (SEQ ID Nr.: 14)
-
Eine
PCR-Reaktionslösung
für 3'-RACE wurde durch
Mischen von 1 μl
50 × Advantage
2 Polymerase Mix (CLONTECH Laboratories, Inc.), 5 μl 10 × Advantage
2 PCR-Puffer angelagert (400 mM Tricine-KOH, 150 mM KOAc, 35 mM
Mg(OAc)2, 37,5 μg/ml BSA, 0,05 % Tween-20, 0,05
% Nonidet-P40), 4 μl
dNTP-Gemisch (jeweils 2,5 mM, TaKaRa Shuzo Co., Ltd.), 1 μl 10 μM Primer
ZF1, 1 μl
10 μM Primer
AP1 (Primer AP1 wurde mit menschlichem Testis-Marathon-Ready-cDNA-Kit
von CLONTECH Laboratories, Inc. zugeführt), 5 μl Matrizen-cDNA (CLONTECH Laboratories,
Inc., menschliche Testis-Marathon-Ready-cDNA) und 33 μl destilliertes Wasser hergestellt.
Die Reaktion wurde unter den Bedingungen durchgeführt: nach
der primären
Denaturierung bei 94 °C
für 60
Sekunden wurde ein Zyklusgruppe bei 94 °C für 30 Sekunden und dann bei
72 °C für 4 Minuten
fünfmal
wiederholt, wurde eine Zyklusgruppe bei 94 °C für 30 Sekunden und dann bei
70 °C für 4 Minuten
fünfmal
wiederholt, und wurde eine Zyklusgruppe bei 94 °C für 30 Sekunden und dann bei
68 °C für 44 Minuten
25 Mal wiederholt.
-
Dann
wurde Nested-PCR unter Verwendung des Reaktionsgemischs aus dem
PCR als eine Matrize durchgeführt.
Die Reaktionslösung
wurde durch Mischen von 1 μl
50 × Advantage
2 Polymerase Mix (CLONTECH), 5 μl
10 × Advantage
2 PCR Puffer angelagert (400 mM Tricine-KOH, 150 mM KOAc, 35 mM
Mg(OAc)2, 37,5 μg/ml BSA, 0,05 % Tween-20, 0,05
% Nonidet-P40), 4 μl
dNTP-Gemisch (jeweils 2,5 mM, TaKaRa Shuzo Co., Ltd.), 1 μl 10 μM Primer
ZF2, 1 μl
10 μM Primer
AP2 (Primer AP2 wurde mit menschlichem Testis-Marathon- Ready-cDNA-Kit von
CLONTECH Laboratories, Inc. zugeführt), 5 μl Matrizen-DNA (50fache Verdünnung des
PCR-Reaktionsgemisches) und 33 μl
destilliertes Wasser hergestellt. Die Reaktion wurde unter den Bedingungen
durchgeführt:
nach der primären
Denaturierung bei 94 °C
für 60
Sekunden wurde eine Zyklusgruppe bei 94 °C für 30 Sekunden und dann bei
72 °C für 4 Minuten
fünfmal
wiederholt, wurde eine Zyklusgruppe bei 94 °C für 30 Sekunden und dann bei
70 °C für 4 Minuten
fünfmal
wiederholt, und eine Zyklusgruppe bei 94 °C für 30 Sekunden und dann bei
68 °C für 44 Minuten
25 Mal wiederholt.
-
Außerdem wurde
zweites Nested-PCR unter Verwendung der Reaktionslösung des
PCR als eine Matrize durchgeführt.
Die Reaktionslösung
wurde durch Mischen von 1 μl
50 × Advantage
2 Polymerase Mix (CLONTECH Laboratories, Inc.), 5 μl 10 × Advantage
2 PCR Puffer angelagert (400 mM Tricine-KOH, 150 mM KOAc, 35 mM
Mg(OAc)2, 37,5 μg/ml BSA, 0,05 % Tween-20, 0,05
% Nonidet-P40), 4 μl
dNTP-Gemisch (jeweils 2,5 mM, TaKaRa Shuzo Co., Ltd.), 1 μl 10 μM Primer
ZF3, 1 μl
10 μl Primer
AP2 (Primer AP2 wurde mit menschlichem Testis-Marathon-Ready-DNA-Kit
von CLONTECH Laboratories, Inc. zugeführt), 5 μl Matrizen-cDNA (50fache Verdünnung des
PCR-Reaktionsgemisches) und 33 μl
destilliertes Wasser hergestellt. Die Reaktion wurde unter den Bedingungen
durchgeführt:
nach der primären
Denaturierung bei 94 °C
für 60
Sekunden wurde eine Zyklusgruppe bei 94 °C für 30 Sekunden und dann bei
72 °C für 4 Minuten
fünfmal
wiederholt, wurde eine Zyklusgruppe bei 94 °C für 30 Sekunden und dann bei
70 °C für 4 Minuten
fünfmal
wiederholt, und wurde eine Zyklusgruppe bei 94 °C für 30 Sekunden und dann bei
68 °C für 44 Minuten
25 Mal wiederholt. Das erhaltene DNA-Fragment wurde unter Verwendung
von TOPO TA Cloning Kit (Invitrogen, Inc.) gemäß dem Protokoll, das in dem
beiliegenden Handbuch beschrieben ist, geklont. Die Basensequenz
der geklonten DNA wurde unter Verwendung eines ABI377DNA-Sequenzer
dekodiert, um die 3'-Ende-Sequenz
(SEQ ID NR. 15) zu identifizieren.
-
Basierend
auf der Basensequenz, die durch SEQ ID NR. 15 dargestellt ist, und
der Informationen aus EST (X40467) wurden Primer ZAQL-CF (SEQ ID
NR. 16) und Primer ZAQL-XR1
(SEQ ID NR. 17) hergestellt. Unter Verwendung von menschlicher Testis-Marathon-Ready-cDNA (CLONTECH
Laboratories, Inc.) als Matrize wurde PCR unter Verwendung von Primern
ZAQL-CF und ZQAL-XR1 durchgeführt.
ZAQL-CF:
5'-CCACCATGAGAGGTGCCACG-3' (SEQ ID Nr.: 16)
ZAQL-XR1:
5'-CTCGAGCTCAGGAAAAGGATGGTG-3' (SEQ ID Nr.: 17)
-
Die
Reaktionslösung
wurde durch Mischen von 1 μl
PfuTurbo-DNA-Polymerase (Stratagene, Inc.), 5 μl 10 × PCR Puffer angelagert, 4 μl 2,5 mM
dNTP-Gemisch, jeweils 2,5 μl
10 μM Primer
ZAQL-CF und ZAQL-XR1, 5 μl
Matrizen-DNA und 30 μl
destilliertem Wasser hergestellt. Die Reaktion wurde unter den Bedingungen
durchgeführt:
nach der primären
Denaturierung bei 95 °C
für 1 Minute
wurde eine Zyklusgruppe bei 95 °C
für 1 Minute
und dann bei 72 °C
für 1 Minute
40 Mal wiederholt, und eine Endextensionsreaktion wurde bei 72 °C für 10 Minuten
durchgeführt.
Das erhaltene DNA-Fragment wurde unter Verwendung von TOPO TA Cloning
Kit (Invitrogen) gemäß dem Protokoll,
das in dem beiliegenden Handbuch beschrieben ist, geklont. Infolge
der Dekodierung der Basensequenzen der geklonten DNA-Fragmente unter
Verwendung des ABI377DNA-Sequenzers wurde herausgefunden, daß die Fragmente
Sequenzen mit 371 Basenpaaren aufwiesen, die durch SEQ ID NR. 18
bzw. SEQ ID NR. 19 dargestellt sind. Das Plasmid, das das DNA-Fragment trägt, das
die Basensequenz enthält,
die durch SEQ ID NR. 18 dargestellt ist, wurde pHMITA genannt, und
das Plasmid, das das DNA-Fragment trägt, das die Basensequenz enthält, die
durch SEQ ID NR. 19 dargestellt ist, wurde pHMITG genannt.
-
Escherichia
coli wurde unter Verwendung der Plasmide pHMITA und pHMITG transfektiert,
und die erhaltenen Transformanten wurden Escherichia coli TOP10/pHMITA
bzw. Escherichia coli TOP 10/pHMITG genannt.
-
Die
Analyse der Basensequenzen der DNA-Fragmente offenbarten, daß das DNA-Fragment,
das durch SEQ ID NR. 18 dargestellt ist, die DNA aufwies (SEQ ID
NR. 28), die das menschentypische ZAQ-Ligandenpräkursorpeptid kodiert (Typ A,
105 Aminosäurereste),
dargestellt durch SEQ ID NR. 22, und daß das DNA-Fragment, das durch
SEQ ID NR. 19 dargestellt ist, die DNA aufwies (SEQ ID NR. 29),
die das menschentypische ZAQ-Ligandenpräkursorpeptid kodiert (Typ G,
105 Aminosäurerest),
dargestellt durch SEQ ID NR. 23.
-
Es
wurde außerdem
herausgefunden, daß die
Basensequenzen, die durch SEQ ID NR. 28 dargestellt sind und durch
SEQ ID NR. 29 dargestellt sind, eine typische Signalsequenz aufwiesen;
die DNA mit der Basensequenz, die durch SEQ ID NR. 28 dargestellt
ist, die DNA auf wies (SEQ ID NR. 26), die aus 258 Basenpaaren besteht
und die das menschentypische reife ZAQ-Ligandenpeptid kodiert (Typ
A, 86 Aminosäurereste), dargestellt
durch SEQ ID NR. 20; die DNA mit der Basensequenz, die durch SEQ
ID NR. 29 dargestellt ist, die DNA aufwies (SEQ ID NR. 27), die
aus 258 Basenpaaren besteht und die das menschentypische reife ZAQ-Ligandenpeptid
kodiert (Typ G, 86 Aminosäurereste),
dargestellt durch SEQ ID NR. 21.
-
BEISPIEL 5
-
Produktion von menschentypischem
ZAQ-Ligandenpeptid in Säugerzellen
(1)
-
(5-1) Konstruktion des
Säugerexpressionsvektors
für menschentypisches
ZAQ-Ligandenpräkursorpeptid
-
Das
Plasmid pHMITG, erhalten in BEISPIEL 4, wurde mit Restriktionsenzymen
EcoRI und XhoI digeriert, wodurch das DNA-Fragment 382 Basenpaaren
(SEQ ID NR. 30), enthaltend cDNA, die das menschentypische ZAQ-Ligandenpräkursorpeptid
kodiert, exzidiert wurde.
-
Das
heißt,
Plasmid pHMITG wurde mit EcoRI und XhoI digeriert, und das erhaltene
DNA-Fragment wurde
auf 1,5 % Agarosegel der Elektrophorese unterworfen. Das Gelsegment
der Bande mit 382 Basenpaaren, eingefärbt mit Cybergreen, wurde mit
einer Rasierklinge herausgeschnitten. Aus dem obigen Gelsegment wurde
das DNA-Fragment unter Verwendung von Gene Clean Spin DNA Extraction
Kit (BIO 101, Inc.) gewonnen. Gemäß dem Standardverfahren wurde
das erhaltene DNA-Fragment in den Säugerzellenexpressionsvektor
pCAN618 (11), der CMV-IE-Enhancer und
Huhn-beta-Actin-Promotor als Expressionspromotor an der Spaltungsstelle
trägt,
durch Restriktionsenzyme EcoRI und XhoI geklont. Die Basensequenz
des geklonten DNA-Fragmentes wurde durch das obige Verfahren dekodiert.
Daher wurde das DNA-Fragment identifiziert und wies die Basensequenz
auf, die durch SEQ ID NR. 30 dargestellt ist. Dieser Säugerzellenexpressionsvektor
mit der DNA, die das menschentypische ZAQ-Ligandenpräkursorpeptid
kodiert, wurde pCANZAQLg2 genannt.
-
(5-2) Einführung von
Expressionsvektor in COS7-Zellen
-
COS7-Zellen
wurden von ATCC bezogen, und die Zellen, die durch DMEM-Medium (ergänzt mit
10 % FBS) in Subkultur angelegt wurden, wurden verwendet. Unter
Verwendung des DMEM-Mediums wurden die COS7-Zellen bei einer Population
von 1,5 × 106 Zel len/Schale auf einer 10-cm-Petrischale
inokuliert, gefolgt von Inkubation bei 37 °C unter 5 % CO2 über Nacht.
Zu 2 μg
des Expressionsplasmids von menschentypischem ZAQ-Ligandenpräkursorpeptid
(gelöst
in 2 μl
TE-Puffer) (pCANZAQLg2) wurden 298 μl Puffer EC(Effectene-Transfektionsreagens,
QIAGEN) zugegeben, und 16 μl
Enhancer wurden außerdem
zu dem Gemisch zugegeben. Nach dem Mischen für eine Sekunde wurde das Gemisch
bei Raumtemperatur für
3 Minuten stehengelassen. Dann wurden 60 μl Effectene-Transfektionsreagens weiterhin zu dem
Gemisch zugegeben. Nach dem Mischen für 10 Sekunden wurde das Gemisch
bei Raumtemperatur für
10 Minuten stehengelassen. Nachdem der Überstand aus den Zellen, die
am Tag zuvor inokuliert wurden, entfernt wurde, wurden die Zellen einmal
mit 10 ml DMEM-Medium gewaschen, und 9 ml DMEM-Medium wurden zugegeben.
Ein Milliliter des DMEM-Mediums wurde zu der Plasmidlösung zugegeben,
und nach dem Mischen wurde das Gemisch tropfenweise zu den Zellen
zugegeben. Nach dem Mischen des gesamten Gemisches wurden die Zellen
bei 37 °C
unter 5 % CO2 über Nacht kultiviert. Die Zellen
wurden zweimal mit 10 ml DMEM-Medium gewaschen, und 10 ml des DMEM-Mediums
wurden zugegeben. Die Zellen wurden in einem Inkubator bei 37 °C unter 5
% CO2 über
Nacht kultiviert. Nach 2 Tagen wurde der Kulturüberstand gewonnen.
-
(5-3) Teilreinigung von
menschentypischem ZAQ-Ligandenpräkursorpeptid
aus dem Kulturüberstand
von Expressions-COS7-Zellen
-
(5-3-1) Herstellung des
Kulturüberstandes
von menschentypisches ZAQ-Ligandenpräkursorpeptid-exprimierenden
COS7-Zellen
-
Der
Kulturüberstand
von menschentypisches ZAQ-Ligandenpräkursorpeptid-exprimierenden COS7-Zellen
wurde gewonnen, und das Extrakt wurde durch die folgende Verfahrensweise
hergestellt. Zunächst
wurden 1,1 ml Essigsäure
tropfenweise zu dem Zellkulturüberstand
(etwa 18,5 ml) zugegeben, wodurch die Endkonzentration auf 1 M eingestellt
wurde, und das Gemisch wurde für
eine Stunde gerührt.
Aceton wurde in 2fachem Volumen des Gemisches zugegeben, welches
dann für
30 Minuten bei 4 °C
gerührt
wurde. Als nächstes
wurde das Gemisch bei 15.000 U/min für 30 Minuten unter Verwendung
einer Hochgeschwindigkeitszentrifuge zentrifugiert (CR26H, Model
23 Rotor: Hitachi, Ltd), wodurch der Überstand erhalten wurde. Der
erhaltene Überstand
wurde in einem Verdampfer gegeben, um Aceton zu entfernen, und dann
mit einem Gefriertrockner lyophilisiert (12EL; VirTis).
-
(5-3-2) Sephadex-G50-Gelchromatographie
und SepPak-Säulenchromatographie
des Kulturüberstandes
von menschentypisches ZAQ-Ligandenpräkursorpeptid-exprimierenden
COS7-Zellen
-
Die
lyophilisierten Pulver, erhalten in (5-3-1) oben, wurden in 2 ml
1 M Essigsäure
gelöst,
und die Lösung
wurde auf Sephadex-G15-Säule
(3 cm × 35
ml, Pharmacia Biotech) adsorbiert, die mit 1 M Essigsäure äquilibriert
wurde. Dann wurden 1 M Essigsäure
durch die Säule
geführt,
und jeweils 5 ml des Eluats wurden entnommen, wobei jeder Fraktion
eine Fraktionsnummer verliehen wurde. Jede Fraktion wurde mit einem
Gefriertrockner lyophilisiert (12EL; VirTis).
-
Eine
SepPak-C18-Sg-Säule
(10 ml) wurde mit Methanol gequollen und mit 0,1 % Trifluoressigsäure/destilliertem
Wasser äquilibriert.
Die lyophilisierten Produkte von Fraktion Nr. 1 bis 16, entnommen
aus den Fraktionen, erhalten durch Sephadex-G50-Gelchromatographie,
wurden zusammengefaßt
und in 3 ml Trifluoressigsäure/destilliertem
Wasser gelöst,
und die Lösung
wurde auf die SepPak-C18-5g-Säule
geladen. Nach dem Waschen mit 24 ml 0,1 % Trifluoressigsäure/destilliertem
Wasser wurde die Säule
gewaschen und mit 20 ml 0,1 % Trifluoressigsäure/60 % Acetonitril eluiert.
Das erhaltene Eluat wurde auf Savant aufgebracht.
-
(5-3-3) Reinigung von
Super ODS Umkehphasen-Hochleistungsflüssigchromatographie
-
Elutionsmittel
A (0,1 % Trifluoressigsäure/destilliertes
Wasser) wurde durch die Säule
zur TSKgel Super ODS Umkehrphasen-Hochleistungsflüssigchromatographie
(Toso, 0,46 cm × 10
cm) bei einer Fließgeschwindigkeit
von 1 ml/min bei 40 °C
geführt,
um die Säule
zu äquilibrieren.
Nachdem die SepPak-C18-5g-Säulenfraktion,
erhalten in (5-3-2), auf Savant aufgebracht wurde, wurde die Fraktion
auf Super-ODS-Umkehrphasen-Hochleistungsflüssigchromatographie geladen.
Dann wurde die Elution durch Erhöhen
mit einem linearen Gradienten von 100 Vol.-% Elutionsmittel A (0,1
% Trifluoressigsäure/destilliertes
Wasser)/0 Vol.-% Elutionsmittel B (0,1 % Trifluoressigsäure/100
% Acetonitril) auf 0 Vol.-% Elutionsmittel A/100 Vol.-% Elutionsmittel
B bei einer Fließgeschwindigkeit
von 1 ml/min über
60 Minuten durchgeführt,
wodurch das Eluat gewonnen wurde.
-
Das
Eluat wurde mit jeweils 1 ml fraktioniert, wobei jeder Fraktion
eine Fraktionsnummer verliehen wurde. Die Gesamtmenge der fraktionierten
Fraktionen wurde mit einem Gefriertrockner lyophilisiert (12EL; VirTis).
Die lyophilisierten Produkte wurden in 150 μl H/HBSS, ergänzt mit
2,5 mM Probenecid und 0,2 % BSA, gelöst. Unter Verwendung dieser
Lösung
wurde die Wirkung des Aktivierens des Rezeptors für ZAQC-B1-Zellen
durch das nachstehend beschriebene Verfahren (5-3-4) analysiert.
-
(5-3-4) Messung der die
intrazelluläre
Ca2+-Ionenkonzentration erhöhenden Aktivität durch
FLIPR
-
Unter
Verwendung von FLIPR wurden die Proben, die oben in (5-3-4) erhalten
wurden, hinsichtlich der die intrazelluläre Ca2+-Ionenkonzentration
erhöhenden
Aktivität
in ZAQ-Expressionszellen
(ZAQC-B1), erhalten in BEISPIEL 3 (3-5), analysiert. Zur Kontrolle
wurden hOT7T175-Expressionszellen (hOT7T175-16; beschrieben in WO
00/24890) verwendet.
-
Die
ZAQC-B1-Zellen und hOT7T175-Zellen wurden in DMEM, ergänzt mit
10 % dialysiertem fetalem Rinderserum (hierin nachstehend als dFBS
bezeichnet), in Subkultur angelegt und zur Verwendung bereitgestellt.
Die ZAQC-B1-Zellen und hOT7T175-Zellen wurden jeweils in dem Medium
(10 % dFBS-DMEM) suspendiert, wodurch ihre Konzentration auf 15 × 104 Zellen/ml eingestellt wurde. Unter Verwendung
einer Dispensierpipette wurden jeweils 200 μl der Zellsuspension (3,0 × 104 Zellen/200 μl/Loch) in jedem Loch inokuliert (Black
Plate Clear Bottom, Coster). Nach der Inkubation bei 37 °C unter 5
% CO2 für
einen Tag wurden die Zellen verwendet (hierin nachstehend als die
Zellplatte bezeichnet). Einundzwanzig Milliliter von H/HBSS (9,8 g
HANKS', 0,35 g Natriumhydrogencarbonat
und 4,77 g HEPES; eingestellt auf pH 7,4 mit Natriumhydroxid, gefolgt
von Sterilisation durch einen Sterilisationsfilter), 210 μl 250 mM
Probenecid und 210 μl
fetales Rinderserum (FBS) wurden gemischt. Ebenso wurden 2 Phiolen
(50 μg)
Fluo 3-AM in 42 μl
Dimethylsulfoxid und 42 μl
20%ige Pluronsäure
gelöst,
und die resultierende Lösung
wurde zu dem obigen H/HBSS-Probenecid-FBS-Gemisch zugegeben. Nach
dem Mischen wurde das Medium von der Zellplatte entfernt, und jeweils 100 μl des Gemisches
wurden in jedes Loch der Zellplatte unter Verwendung einer 8-Kanalpipette
dispensiert. Die Zellplatte wurde dann bei 37 °C unter 5 % CO2 für eine Stunde
inkubiert (Farbstoffladung). Für
die Assayproben, erhalten in dem oben beschriebenen BEISPIEL (5-3-3),
wurden 150 μl
H/HBSS, enthaltend 2,5 mM Probenecid und 0,2 % FBS, zu jeder Fraktion
zur Auflösung
zugegeben. Die Lösung
wurde dann zu einer 96-Lochplatte (V-Bottom Plate, Coster) für FLIPR übertragen
(hierin nachstehend als Probenplatte bezeichnet). Nach Beendigung
der Farbstoffladung wurde die Zellplatte viermal mit dem Waschpuffer
oder H/HBSS, ergänzt
mit 2,5 mM Probenecid, unter Verwen dung eines Plattenwäschers (Molecular
Devices) gewaschen. Nach dem Waschen wurden 100 μl des Waschpuffers gesichert.
Diese Zellplatte und die Probenplatte wurden auf FLIPR geladen,
um den Assay durchzuführen
(0,05 ml der Probe wurden von der Probenplatte zu der Zellplatte
durch FLIPR übertragen).
Die die intrazelluläre
Ca2+-Ionenkonzentration erhöhende Aktivität, die für ZAQ-B1-Zellen
spezifisch ist, wurde in Fraktion Nr. 48 bis 68 beobachtet. Daraus
wurde herausgefunden, daß die
Zielkomponente mit der Funktion zum Aktivieren des Rezeptors für ZAQC-B1-Zellen,
d. h., die ZAQ-aktivierende Komponente, in Fraktion Nr. 48 bis 68
eluiert wurde.
-
BEISPIEL 6
-
Produktion von menschentypischem
ZAQ-Ligandenpeptid in Säugerzellen
(2)
-
(6-1) Herstellung des
Kulturüberstandes
-
Wie
in BEISPIEL 5 beschrieben, wurde das Expressionsplasmid von menschentypischem
ZAQ-Ligandenpräkursorpeptid
(pCANZAQLg2) in COS7-Zellen eingeführt. Das heißt, COS7-Zellen
wurden bei einer Population von 3,0 × 106 Zellen/Schale
auf einer 15-cm-Petrischale
inokuliert, gefolgt von Inkubation bei 37 °C unter 5 % CO2 über Nacht.
Nachdem 600 μl
Puffer EC (Effectene-Transfektionsreagens, QIAGEN) zu 4 μg des menschentypischen
ZAQ-Ligandenpräkursorpeptid-Expressionsplasmids
(pCANZAQLg2) (gelöst
in 4 μl TE-Puffer)
zugegeben wurde, wurden 32 μl
Enhancer weiter zugegeben. Nach dem Mischen für eine Sekunde wurde das Gemisch
für 3 Minuten
bei Raumtemperatur stehengelassen. Außerdem wurden 120 μl Effectene-Transfektionsreagens
zu dem Gemisch zugegeben. Nach dem Mischen für 10 Sekunden wurde das Gemisch
für 10
Minuten bei Raumtemperatur stehengelassen. Der Überstand wurde aus den Zellen,
die am Tag zuvor inokuliert wurden, entfernt, und die Zellen wurden
einmal mit 10 ml DMEM gewaschen, und 30 ml DMEM wurden zugegeben.
Zu der Plasmidlösung
wurde 1 ml DMEM zugegeben, und nach dem Mischen wurde das Gemisch
tropfenweise zu den Zellen zugegeben. Nach dem Mischen des gesamten
Gemisches wurden die Zellen bei 37 °C unter 5 % CO2 über Nacht
kultiviert. Die Zellen wurden einmal mit 10 ml DMEM gewaschen, und
20 ml DMEM wurden zugegeben. Die Zellen wurden in einem Inkubator
bei 37 °C
unter 5 % CO2 über Nacht inkubiert. Am nächsten Tag
wurde der Kulturüberstand
gesammelt, und 20 ml DMEM wurden zu dem Sy stem zugegeben. Das Gemisch
wurde in einem Inkubator bei 37 °C
unter 5 % CO2 über Nacht kultiviert, wodurch
der Kulturüberstand
gewonnen wurde.
-
(6-2) Reinigung von menschentypischem
ZAQ-Ligandenpeptid aus dem Kulturüberstand
-
Unter
Verwendung der in (6-1) oben beschriebenen Verfahrensweise wurde
das Kulturmedium aus 80 Petrischalen von 15 cm gewonnen. Essigsäure wurde
zu dem Medium zugegeben, wodurch die Endkonzentration 1 M betrug.
Nach 1 Stunde Rühren
wurde Aceton in einem zweifachen Volumen der Lösung zugegeben, wodurch Proteine
ausfielen. Die Lösung
wurde für
30 Minuten bei 4 °C
gerührt.
Dann wurde die Lösung
bei 10.000 U/min für
30 Minuten mit einer Hochgeschwindigkeitszentrifuge zentrifugiert
(CR26H RR10A-Typ Rotor: Hitachi System Engineering Co., Ltd.), wodurch
ein Überstand
erhalten wurde. Der erhaltene Überstand wurde
auf einen Verdampfer aufgebracht, um Aceton zu entfernen, und durch
eine Umkehrphasensäule
(Waters C18, 100 g) geführt,
die zuvor mit 0,1 % Trifluoressigsäure/destilliertes Wasser äquilibriert
wurde. Nach dem die Säule
mit 1.000 ml 0,1 % Trifluoressigsäure/destilliertem Wasser und
dann mit 1.000 ml 0,1 % Trifluoressigsäure/20 % Acetonitril gewaschen
wurde, wurde das Peptid mit 1.000 ml 0,1 % Trifluoressigsäure/60 %
Acetonitril eluiert. Das erhaltene Eluat wurde auf einen Verdampfer
aufgebracht, und mit einem Gefriertrockner lyophilisiert (12EL;
VirTis).
-
Elutionsmittel
A (0,1 % Trifluoressigsäure/destilliertes
Wasser) wurde durch die Säule
zur TSKgel-ODS80TM-Umkehrphasen-Hochleistungsflüssigchromatographie (Toso,
21,5 min × 30
cm) bei einer Fließgeschwindigkeit
von 4 ml/min bei 40 °C
geführt,
wodurch die Säule äquilibriert
wurde. Nachdem die erhaltenen lyophilisierten Pulver in Elutionsmittel
A gelöst
wurden, wurde die Lösung
auf die ODS80TM-Säule geladen,
wurde das Peptid durch Erhöhen
mit einem linearen Gradienten von 60 Vol.-% Elutionsmittel A (0,1 %
Trifluoressigsäure/destilliertes
Wasser)/40 Vol.-% Elutionsmittel B (0,1 % Trifluoressigsäure/60 %
Acetonitril) auf 0 Vol.-% Elutionsmittel A/100 Vol.-% Elutionsmittel
B bei einer Fließgeschwindigkeit
von 1 ml/min über 120
Minuten eluiert.
-
Das
Eluat wurde mit jeweils 8 ml fraktioniert, wobei jeder Fraktion
eine Fraktionsnummer verliehen wurde. Aus den erhaltenen Fraktionen
wurden jeweils 50 μl
entnommen und mit einem Gefriertrockner lyophilisiert (12EL; VirTis).
Die lyophilisierten Produkte wurden in 200 ul H/HBSS, ergänzt mit
2,5 mM Probenecid und 0,2 % BSA, gelöst. Unter Verwendung dieser
Lösung
wurde die Wirkung zum Aktivieren des Rezeptors für ZAQC-B1-Zellen durch das
nachstehend beschriebene Testverfahren (5-3-4) analysiert. Die Ergebnisse
offenbarten, daß die
Zielkomponente mit der Wirkung zum Aktivieren des Rezeptors für ZAQC-B1-Zellen, d. h., die
ZAQ-aktivierende Komponente, in Fraktion Nr. 32 eluiert wurde.
-
Elutionsmittel
A (10 mM Ammoniumformiat/10 % Acetonitril) wurde durch die Säule zur TSKgel-CM-2SW-Ionenaustausch-Hochleistungsflüssigchromatographie
(Toso, 4,6 mm × 25
cm) bei einer Fließgeschwindigkeit
von 1 ml/min bei 25 °C
geführt,
wodurch die Säule äquilibriert
wurde. Die oben beschriebene Fraktion Nr. 32 wurde auf die CM-2SW-Säule geladen,
und das Peptid wurde mit einem linearen Gradienten durch Erhöhen von
100 Vol.-% Elutionsmittel A (10 mM Ammoniumformiat/10 % Acetonitril)/0
Vol.-% Elutionsmittel B (1000 mM Ammoniumformiat/10 % Acetonitril)
auf 0 Vol.-% Elutionsmittel A/100 Vol.-% Elutionsmittel B bei einer
Fließgeschwindigkeit
von 1 ml/min über
60 Minuten eluiert.
-
Das
Eluat wurde mit jeweils 1 ml fraktioniert, wobei jeder Fraktion
eine Fraktionsnummer verliehen wurde. Aus den erhaltenen Fraktionen
wurden 1,5 μl
entnommen und mit 200 μl
H/HBSS, ergänzt
mit 2,5 mM Probenecid und 0,2 % BSA, verdünnt. Diese Lösung wurde
verwendet, um die Funktion zum Aktivieren des Rezeptors für ZAQC-B1-Zellen
durch das oben beschriebene Testverfahren (5-3-4) zu analysieren.
Die Ergebnisse offenbarten, daß die
Zielkomponente mit der Funktion zum Aktivieren des Rezeptors für ZAQC-B1-Zellen,
d. h., die ZAQ-aktivierende Komponente, in Fraktion Nr. 56 und 57
eluiert wurde.
-
Elutionsmittel
A (0,1 % Trifluoressigsäure/destilliertes
Wasser) wurde durch die Säule
zur TSKgel-Super-Phenyl-Umkehrphasen-Hochleistungsflüssigchromatographie
(Toso, 4,6 mm × 10
cm) bei einer Fließgeschwindigkeit
von 1 ml/min bei 40 °C
geführt,
um die Säule
zu äquilibrieren.
Die obigen Fraktionen Nr. 56 und 57 wurden auf die Super-Phenyl-Säule geladen,
und das Peptid wurde durch Erhöhen
mit einem linearen Gradienten von 70 Vol.-% Elutionsmittel A (0,1
% Trifluoressigsäure/destilliertem
Wasser)/30 Vol.-% Elutionsmittel B (0,1 % Trifluoressigsäure/60 %
Acetonitril) auf 50 Vol.-% Elutionsmittel A/50 Vol.-% Elutionsmittel
B über
60 Minuten bei einer Fließgeschwindigkeit
von 1 ml/min eluiert.
-
Das
Eluat wurde mit jeweils 1 ml fraktioniert, wobei jeder Fraktion
eine Fraktionsnummer verliehen wurde, und 1,5 μl des Eluats wurden aus den
erhaltenen Fraktionen entnommen.
-
Das
Eluat wurde mit 200 μl
H/HBSS, ergänzt
mit 2,5 mM Probenecid und 0,2 % BSA, verdünnt. Diese Lösung wurde
verwendet, um die Funktion zum Aktivieren des Rezeptors für ZAQC-B1-Zellen
durch das oben beschriebene Testverfahren (5-3-4) zu analysieren.
Die Ergebnisse offenbarten, daß die
Zielkomponenten mit Funktion zum Aktivieren des Rezeptors für ZAQC-B1-Zellen,
nämlich
die ZAQ-aktivierende Komponente, in Fraktion Nr. 54, 55 und 56 eluiert
wurde. Die Aktivität
stimmte mit dem einzelnen UV-Absorptionspeak überein, was so interpretiert
wurde, daß die
aktivierende Komponente genug zur Homogenität gereinigt wurde.
-
Die
gereinigte ZAQ-aktivierende Komponente wurde lyophilisiert, und
das lyophilisierte Produkt wurde in Lösungsmittel DMSO (Dimethylsulfoxid)
gelöst.
Ein aliquoter Teil der Lösung
wurde für
das N-terminale Aminosäuresequenzieren
unter Verwendung eines Proteinsequenzers bereitgestellt (Perkin-Elmer,
Inc., PE Biosystems Procise 491cLC). Infolgedessen konnten 9 Reste
von den Aminosäureresten
von dem N-Terminus bis zu dem 10. Aminosäurerest identifiziert werden
(Ala Val Ile Thr Gly Ala Xaa Glu Arg Asp (SEQ ID NR. 31; Xaa ist
ein nicht-identifizierter Rest)). Die erhaltene Aminosäuresequenz
stimmte mit der N-terminalen Aminosäuresequenz des vorhergesehenen
menschentypischen reifen ZAQ-Ligandenpeptids überein.
Ebenso wurde die Massenspektrometrie für die gereinigte Probe der
ZAQ-aktivierenden Komponente unter Verwendung einer Finnigan LCQ
LC/MC-Vorrichtung
(Thermoquest, San Jose, CA) gemäß dem Elektrosprayionisationsverfahren
durchgeführt,
wobei das Molekulargewicht 9657,6 betrug. Dieses Ergebnis stimmte
mit dem berechneten Wert von 9657,3 für menschentypisches reifes
ZAQ-Ligandenpeptid mit 86 Resten überein, bei denen alle 10 Cysteinreste
Disulfidbindungen bildeten. Es wurde bestätigt, das die gereinigte Probe
der ZAQ-aktivierenden Komponente reifes menschentypisches ZAQ-Ligandenpeptid
mit der Aminosäuresequenz,
die durch SEQ ID NR. 21 dargestellt, aufwies.
-
(6-3) Assay für die ZAQ-aktivierende
Funktion von gereinigtem menschentypischen ZAQ-Ligandenpeptid
-
Die
Funktion zum Aktivieren des Rezeptors für ZAQC-B1-Zellen durch das
menschentypische reife ZAQ-Ligandenpeptid, gereinigt in (6-2) oben,
wurde durch das oben in (5-3-4) beschriebene Testverfahren analysiert.
Die Ergebnisse offenbarten, daß das
menschentypische reife ZAQ-Ligandenpeptid eine Erhöhung der
intrazellulären
Calciumionenkonzentrati on in den ZAQ-exprimierten CHO-Zellen (ZAQC-B1-Zellen)
konzentrationsabhängig
induzierte. Der EC50-Wert betrug 96 pM,
und es wurde entdeckt, daß menschentypisches reifes
ZAQ-Ligandenpeptid eine sehr wirksame Agonistenaktivität zeigte.
Die Ergebnisse werden in 10 gezeigt.
-
BEISPIEL 7
-
Produktion von menschentypischen
ZAQ-Ligandenpeptid in Säugerzellen
(3)
-
(7-1) Herstellung einer
CHO-Zellinie, die das menschentypische ZAQ-Ligandenpeptid stabil
exprimieren kann
-
Unter
Verwendung von Plasmid pHMITG als eine Matrize, beschrieben in BEISPIEL
4, wurde menschentypische ZAQ-Liganden-cDNA durch PCR unter Verwendung
der folgenden Primer amplifiziert:
5' GTCGACCACCATGAGAGGTGCCACGC 3' (SEQ ID Nr.: 32)
5' ACTAGTCGCAGAACTGGTAGGTATGG
3' (SEQ ID Nr.:
33)
und zu pCR-Blunt-II-Vektor (Invitrogen, Inc.) geklont.
Die Insert-cDNA wurde aus dem erhaltenen Klon mit einer korrekten
Sequenz unter Verwendung von Restriktionsenzymen SalI und SpeI exzidiert,
und in pAKKO1.1H-Expressionsvektor eingeführt. Das Plasmid wurde zu CHO/dhFr–-Zellen
(American Type Culture Collection) durch die in (3-5) oben beschriebene
Verfahrensweise unter Verwendung von CellPhect Transfection Kit
transfektiert (Amersham Pharmacia Biotech). Der Kulturüberstand
wurde aus mehreren Klonen des transfektierten Stamms gewonnen, und
die ZAQ-Ligandenaktivität,
die die intrazelluläre
Ca2+-Ionenkonzentration in den ZAQC-B1-Zellen
erhöhen
kann, wurde durch das in (3-5) beschriebene Testverfahren analysiert. Daher
wurde ZAQL-exprimierter CHO-Zellenklon Nr. 4, der den ZAQ-Liganden
stark exprimieren kann, gescreent.
-
(7-2) Herstellung von
serumfreiem Kulturüberstand
von menschentypischem ZAQ-Ligand ZAOL-1
-
In
Dulbecco's Modifizierten
Eagle-Medium (DMEM) (Nissui Pharmaceutical Co., Ltd.), ergänzt mit
10 % dialysiertem fetalem Rinderserum (JRH BIOSCIENCES), 1 mM MEM
nicht-essentieller
Aminosäurelösung, 100
Einheiten/ml Penicillin und 100 μg/ml
Streptomyzin, wurde ZAQL-1-exprimierter CHO-Zellenklon Nr. 4 kultiviert,
um unter Verwendung von 4 Single Trays (Nunc) konfluierend zu werden,
und mit Trypsin zur Dispersion behandelt. Die Dispersion wurde zentrifugiert,
wodurch der Klon gewonnen wurde. Die Zellen, die einer der obigen
Single Trays entsprechen, wurden in 1,5 l des Mediums suspendiert,
und in Cell Factories 10 (Nunc) inokuliert, gefolgt von Inkubieren
von 4 Cell Factories 10 bei 37 °C
für 3 Tage
unter 5 % CO2. Nach dem der Kulturüberstand
entfernt wurde, wurden die Zellen in einer der Cell Factories 10
mit 1 l oben beschriebenem H/HBSS gewaschen. Nachdem H/HBSS entfernt
wurde, wurde serumfreies Medium (Dulbecco's Modifiziertes Eagle-Medium, ergänzt mit
1 mM nicht-essentieller Aminosäurelösung, 100
Einheiten/ml Penicillin und 100 μg/ml
Streptomyzin) in 2 l pro Cell Factory 10 zugegeben, gefolgt von
Inkubation für
weitere 2 Tage. Der gewonnene Kulturüberstand wurde bei 1.000 U/min
für 10
Minuten unter Verwendung einer Hitachi-Hochgeschwindigkeitszentrifuge
zentrifugiert und durch Gaze filtriert, wodurch ein klarer Überstand
erhalten wurde. Essigsäure
wurde zu dem Überstand
in einer Endkonzentration von 1 M zugegeben.
-
(7-3) Grobfraktionierung
von ZAQL-1-Expressions-CHO-Zellen-Serum-freiem Kulturüberstand
durch Wakosil-II 5C 18HG Prep Umkehrphasen-Hochleistungsflüssigchromatographie
-
PrepC18
(Waters), gepackt mit Octadecyl-fixiertem Kieselgel, quoll in Methanol
und wurde in eine Säule
aus Glas gepackt (50 mm × 100
mm). Dann wurde das Extrakt, hergestellt in (6-2), auf eine Säule geladen,
die mit 1 M Essigsäure äquilibriert
wurde. Dann wurde die Säule
mit 800 ml von 1 M Essigsäure
gewaschen. Dann wurden 1000 ml von 60 % Acetonitril/0,1 % Trifluoressigsäure durch
die Säule
geführt,
um die rohe Zielpeptidkomponente zu eluieren. Das erhaltene Eluat
wurde unter Verwendung eines Verdampfers konzentriert und mit einem
Gefriertrockner lyophilisiert (12EL; VirTis).
-
(7-4) Trennung durch Wakosil-II
5C18HG Prep Umkehrphasen-Hochleistungsflüssigchromatographie
-
Mittels
Führen
von 91,7 Vol.-% Elutionsmittel A (0,1 % Trifluoressigsäure/destilliertes
Wasser)/8,3 Vol.-% Elutionsmittel B (0,1 % Trifluoressigsäure/60 %
Acetonitril) durch eine Säule
zur Wakosil-II 5C18HG Prep Umkehrphasen-Hochleistungsflüssigchromatographie
(Wako Pure Chemical Industries, 20 mm × 250 mm) bei 40 °C bei einer
Fließgeschwindigkeit
von 5 ml/min wurde die Säule äquilibriert.
Das oben in (7-3) erhaltene lyophilisierte Produkt wurde dem Chromatographieverfahren
unterzogen. Das heißt,
36 ml von 1 M Essigsäure
wurden zugegeben, wodurch das lyophilisierte Produkt gelöst wurde.
Nachdem die Lösung
zentrifugiert wurde, wurde ein aliquoter Teil von einem Drittel
auf die Säule
geladen, und die Konzentration an Elutionsmittel B wurde auf 66,7
Vol.-% Elutionsmittel A/33,3 Vol.-% Elutionsmittel B über eine
Minute erhöht
und dann mit einem linearen Gradienten auf 16,7 Vol.-% Elutionsmittel
A/83,3 Vol.-% Elutionsmittel B über
die nächsten
120 Minuten bei einer Fließgeschwindigkeit
von 5 ml/min erhöht.
Das Eluat wurde mit jeweils 5 ml fraktioniert, wobei jeder Fraktion
eine Fraktionsnummer verliehen wurde, und jeweils 3 μl aus den
erhaltenen Fraktionen wurden mit 150 μl von 0,2 % BSA gemischt, gefolgt
von Lyophilisierung mit einem Gefriertrockner (12EL; VirTis). Das
lyophilisierte Produkt wurde mit 150 μl Assaypuffer zugegeben [H/HBSS
(9,8 g Nissui HANKS 2 (Nissui Pharmaceutical Co., Ltd.), 0,35 g
Natriumhydrogencarbonat und 4,77 g HEPES; eingestellt auf pH 7,4
mit Natriumhydroxid, gefolgt von Sterilisation durch einen Sterilisationsfilter),
ergänzt
mit 2,5 mM Probenecid und 0,1 % CHAPS], wodurch das lyophilisierte
Produkt gelöst
wurde. Unter Verwendung von 50 μl der
Lösung
wurde die ZAQ-B1-Rezeptor-aktivierende Funktion durch das in (3-5)
oben beschriebene Testverfahren analysiert. Die Ergebnisse offenbarten,
daß die
Komponente mit der jeweiligen ZAQ-B1-Rezeptor-aktivierenden Funktion
hauptsächlich
in den Fraktionen Nr. 73 bis 75 eluiert wurde.
-
(7-5) Trennung durch TSKgel-CM-2SW-Ionenaustausch-Hochleistungsflüssigchromatographie
-
Mittels
Führen
von 100 Vol.-% Elutionsmittel A (4 M Ammoniumformiat : destilliertem
Wasser : Acetonitril = 1 : 299 : 100) und 0 Vol.-% Elutionsmittel
B (4 M Ammoniumformiat : destilliertem Wasser : Acetonitril = 1
: 2 : 1) durch eine Säule
zur TSKgel-CM-2SW-Ionenaustausch-Hochleistungsflüssigchromatographie
(Toso, 7,8 mm × 300
mm) bei einer Fließgeschwindigkeit
von 2 ml/min bei 25 °C
wurde die Säule äquilibriert.
In den Fraktionen, erhalten in (7-4) durch Wakosil-II 5C18HG Prep
Umkehrphasen-Hochleistungsflüssigchromatographie,
wurden die Fraktionen Nr. 73 bis 75 lyophilisiert, und das lyophilisierte
Produkt wurde in 4 ml Elutionsmittel A gelöst. Nachdem die Lösung auf
die TSKgel-CM-2SW-Ionenaustauschsäule geladen
wurde, wurde das Eluat durch Erhöhen
mit einem linearen Gradienten auf 25 Vol.-% Elutionsmittel A/75
Vol.-% Elutionsmittel B über
120 Minuten bei einer Fließgeschwindigkeit
von 1 ml/min gewonnen. Das Eluat wurde mit jeweils 2 ml fraktioniert,
wobei jeder Fraktion eine Fraktionsnummer verliehen wurde, und jeweils
10 μl aus
den erhaltenen Fraktionen wurden mit 100 μl von 0,2 % BSA gemischt, gefolgt
von Lyophilisierung mit einem Gefriertrockner (12EL; VirTis). Das
lyophilisierte Produkt wurde mit 100 μl des oben beschriebenen Assaypuffers
zum Lösen zugegeben.
-
Nachdem
die Lösung
auf das 100fache mit demselben Puffer verdünnt wurde, wurde die ZAQ-Rezeptor-aktivierende
Funktion durch das in (3-5) oben beschriebene Testverfahren analysiert.
Die Ergebnisse offenbarten, daß die
Komponente mit der Ziel-ZAQ-Rezeptoraktivierenden Funktion hauptsächlich in
den Fraktionen Nr. 95 bis 100 eluiert wurde.
-
(7-6) Reinigung durch
TSKgel-ODS-80Ts-Umkehrphasen-Hochleistungsflüssigchromatographie
-
Mittels
Führen
von 91,7 Vol.-% Elutionsmittel A (0,1 % Trifluoressigsäure/destilliertes
Wasser) und 8,3 Vol.-% Elutionsmittel B (0,1 % Trifluoressigsäure/60 %
Acetonitril) durch die Säule
zur TSKgel-ODS-80Ts-Umkelrphasen-Hochleistungsflüssigchromatographie (Toso Co.,
Ltd., 4,6 mm × 25
cm) bei einer Fließgeschwindigkeit
von 1 ml/min bei 40 °C
wurde die Säule äquilibriert.
Die lyophilisierten Produkte der Fraktion Nr. 95 bis 100, erhalten
in (7-5) oben, wurden der Chromatographiebehandlung unterzogen.
Das heißt,
die lyophilisierten Produkte wurden in 4 ml von 1 M Essigsäure gelöst, und
die Lösung
wurde auf die Säule
geladen. Die Konzentration an Elutionsmittel B wurde auf 75 Vol.-%
Elutionsmittel A/25 Vol.-% Elutionsmittel B über 1 Minute erhöht und dann
mit einem linearen Gradienten auf 25 Vol.-% Elutionsmittel A/75
Vol.-% Elutionsmittel B über die
nächsten
60 Minuten bei einer Fließgeschwindigkeit
von 1 ml/min erhöht,
wodurch das Eluat gewonnen wurde. Das Eluat wurde mit jeweils 1
ml fraktioniert, wobei jeder Fraktion eine Fraktionsnummer verliehen
wurde. Die ZAQ-Ligandenaktivität
wurde gemäß dem in
(3-5) beschriebenen Testverfahren analysiert, und es wurde bestätigt, daß die Aktivität in der
Fraktion eluiert wurde, die mit dem einzelnen UV-Absorptionspeak übereinstimmt.
Daraus wurde geschlußfolgert,
daß der
ZAQ-Ligand genug zur Homogenität
gereinigt wurde.
-
(7-7) Analyse der Struktur
von gereinigtem ZAQ-Ligandenpeptid
-
Die
folgende Verfahrensweise wurde verwendet, um die Struktur des ZAQ-Ligandenpeptids,
das in dem oben beschriebenen (7-6) erhalten wurde, zu bestimmen.
Die gereinigte hauptsächliche
ZAQ-aktivierende Komponente wurde mit einem Gefriertrockner lyophilisiert
(12EL; VirTis). Das lyophilisierte Produkt wurde in Lösungsmittel
DMSO (Dimethylsulfoxid) gelöst.
Ein aliquoter Teil der Lösung
wurde für
das N-terminale Aminosäuresequenzieren
unter Verwendung eines Proteinsequenzers bereitgestellt (Perkin-Elmer,
Inc., PE Biosystems Procise 491cLC). Infolgedessen wurde die N-terminale
Aminosäuresequenz,
die mit der vorhergesehenen menschentypischen reifen ZAQ-Ligandenpeptidform
(SEQ ID NR. 21) übereinstimmt,
erhalten. Ebenso wurde die Massenspektrometrie gemäß dem Elektronensprühionisationsverfahren
unter Verwendung einer Finnigan LCQ LC/MC-Vorrichtung durchgeführt, und
das Molekulargewicht betrug 9658,0. Dieser gefundene Wert stimmte
gut mit dem berechneten Wert von 9657,3 für menschentypisches reifes
ZAQ-Ligandenpeptid (SEQ ID NR. 21) überein. Es wurde daher bestätigt, daß das Zielpeptid
mit der Aminosäuresequenz,
die durch SEQ ID NR. 21 dargestellt ist, erhalten wurde.
-
BEISPIEL 8
-
Reaktivität von Schlangengift
MIT1 und menschentypischen ZAQ-Ligand mit ZAQ und I5E
-
(8-1) Isolation von Schlangengift
MIT1
-
(8-1-1) Trennung durch
Wakosil-II 5C18HG Prep Umkehrphasen-Hochleistungsflüssigchromatographie
-
Mittels
Führen
von 91,7 Vol.-% Elutionsmittel A (0,1 % Trifluoressigsäure/destilliertes
Wasser)/8,3 Vol.-% Elutionsmittel B (0,1 % Trifluoressigsäure/60 %
Acetonitril) durch eine Säule
zur Wakosil-II 5C18HG Prep Umkehrphasen-Hochleistungsflüssigchromatographie
(Wako Pure Chemical Industries, 20 mm × 250 mm) bei 40 °C bei einer
Fließgeschwindigkeit
von 5 ml/min wurde die Säule äquilibriert.
Zu 50 mg des lyophilisierten Produktes vom Gift der Schwarzen Mamba
(Sigma) wurden 4 ml von 1 M Essigsäure zugegeben, wodurch das
lyophilisierte Produkt gelöst
wurde. Die Lösung
wurde bei 15.000 U/min für
10 Minuten zentrifugiert, und der Überstand wurde der Chromatographie
unterzogen. Nachdem die Probe auf die Säule geladen wurde, wurde die
Konzentration an Elutionsmittel B auf 91,7 Vol.-% Elutionsmittel
A/8,3 Vol.-% Elutionsmittel B über 1
Minute erhöht
und dann mit einem linearen Gradienten auf 33,4 Vol.-% Elutionsmittel
A und 66,6 Vol.-% Elutionsmittel B über 120 Minuten bei einer Fließgeschwindigkeit
von 5 ml/min erhöht.
Das Eluat wurde mit jeweils 10 ml 20 Minuten später fraktioniert, wobei jeder
Fraktion eine Fraktionsnummer verliehen wurde. Aus jeder Fraktion
wurde 1 μl
entnommen, und nach dem Verdünnen
auf das 10.000fache mit dem oben beschriebenen Assaypuffer wurde
die ZAQ-B1-Rezeptor-aktivierende Funktion gemäß dem in (3-5) oben beschriebenen
Testverfahren analysiert. Die Ergebnisse offenbaren, daß die Komponente
mit der ZAQ-B1-Rezeptor-aktivierenden Zielfunktion hauptsächlich in
den Fraktionen Nr. 21 bis 23 eluiert wurde.
-
(8-1-2) Trennung durch
TSKgel-CM-2SW-Ionenaustausch-Hochleistungsflüssigchromatographie
-
Mittels
Führen
von 100 Vol.-% Elutionsmittel A (4 M Ammoniumformiat : destilliertem
Wasser : Acetonitril = 1 : 299 : 100) und 0 Vol.-% Elutionsmittel
B (4 M Ammoniumformiat : destilliertem Wasser : Acetonitril = 1
: 2 : 1) durch eine Säule
zur TSKgel-CM-2SW-Ionenaustausch-Hochleistungsflüssigchromatographie
(Toso, 4,6 mm × 250
mm) bei einer Fließgeschwindigkeit
von 1 ml/min bei 25 °C
wurde die Säule äquilibriert.
Von den durch Wakosil-II 5C18HG Prep Umkehrphasen-Hochleistungsflüssigchromatographie
in (7-1) erhaltenen Faktionen wurden die Fraktionen Nr. 21 bis 23
lyophilisiert, und die lyophilisierten Produkte wurden in 4 ml Elutionsmittel
A gelöst.
Nachdem die Lösung
auf die TSKgel-CM-2SW-Ionenaustauschsäule geladen
wurde, wurde das Eluat durch Erhöhen
mit einem linearen Gradienten auf 0 Vol.-% Elutionsmittel A/100
Vol.-% Elutionsmittel B über
90 Minuten bei einer Fließgeschwindigkeit
von 1 ml/min gewonnen. Das Eluat wurde mit jeweils 1 ml fraktioniert,
wobei jeder Fraktion eine Fraktionsnummer verliehen wurde. Aus jeder
Fraktion wurde 1 μl entnommen,
und nach dem Verdünnen
auf das 10.000fache mit dem oben beschrieben Assaypuffer wurde die ZAQ-B1-Rezeptor-aktivierende
Funktion gemäß dem in
(3-5) oben beschriebenen Testverfahren analysiert. Die Ergebnisse
offenbarten, daß die
Komponente mit der ZAQ-B1-Rezeptor-aktivierenden Zielfunktion hauptsächlich in
den Fraktionen Nr. 50 bis 51 eluiert wurde.
-
(8-1-3) Trennung durch
Vydac238TP3410-Ionenaustausch-Hochleistungsflüssigchromatographie
-
Mittels
Führen
von 91,7 Vol.-% Elutionsmittel A (0,1 % Trifluoressigsäure/destilliertes
Wasser)/8,3 Vol.-% Elutionsmittel B (0,1 % Trifluoressigsäure/60 %
Acetonitril) durch eine Säule
zur Vydac238TP3410-Ionenaustausch-Hochleistungsflüssigchromatographie
(Toso, 4,6 mm × 100
mm) bei einer Fließgeschwindigkeit von
1 ml/min bei 40 °C
wurde die Säule äquilibriert.
Von den Fraktionen, erhalten in (8-1-2) durch TSKgel-CM-2SW-Ionenaustausch-Hochleistungsflüssigchromatographie,
wurden die Fraktionen Nr. 50 bis 51 direkt auf die Säule geladen,
die Konzentration an Elutionsmittel B wurde auf 75 Vol.-% Elutionsmittel
A/25 Vol.-% Elutionsmittel B über
1 Minute erhöht
und dann mit einem linearen Gradienten auf 41,7 Vol.-% Elutionsmittel
A und 58,3 Vol.-% Elutionsmittel B über 75 Minuten bei einer Fließgeschwindigkeit
von 1 ml/min erhöht. Das
Eluat wurde mit jeweils 0,5 ml fraktionert, wobei jeder Fraktion
eine Fraktionsnummer verliehen wurde. Aus jeder Fraktion wurde 1 μl entnommen,
und nach dem Verdünnen
auf das 10.000fache mit dem oben beschriebenen Assaypuffer wurde
die ZAQ-B1-Rezeptor-aktivierende Funktion gemäß dem in (3-5) oben beschriebenen
Testverfahren analysiert. Die Ergebnisse offenbarten, daß die Komponente
mit der ZAQ-B1-Rezeptor-aktivierenden Zielfunktion hauptsächlich in
den Fraktionen Nr. 108 bis 115 eluiert wurde.
-
(8-1-4) Analyse der Struktur
von gereinigtem Schlangengift MIT1
-
Die
folgende Verfahrensweise wurde verwendet, um die Struktur vom Schlangengift
MIT1, erhalten in dem oben beschriebenen (8-1-3), zu bestimmen.
Das gereinigte Haupt-MIT1 wurde mit einem Gefriertrockner (12EL;
VirTis) lyophilisiert. Das lyophilisierte Produkt wurde in Lösungsmittel
DMSO (Dimethylsulfoxid) gelöst. Ein
aliquoter Teil der Lösung
wurde für
das N-terminale Aminosäuresequenzieren
unter Verwendung eines Proteinsequenzers bereitgestellt (Perkin-Elmer,
Inc., PE Biosystems Procise 491cLC). Infolgedessen wurde die N-terminale
Aminosäuresequenz,
die mit dem vorhergesehenen Schlangengift MIT1 (SEQ ID NR. 34) übereinstimmt,
erhalten. Ebenso wurde Massenspektrometrie gemäß dem Elektronensprühionisationsverfahren
unter Verwendung der Finnigan LCQ LC/MC-Vorrichtung durchgeführt, und
das Molekulargewicht betrug 8506,8. Dieser gefundene Wert stimmte
gut mit dem berechneten Wert von 8506,4 für Schlangengift MIT1 überein.
Es wurde daher bestätigt,
daß das
Zielpeptid mit der Aminosäuresequenz,
die durch SEQ ID NR. 34 dargestellt ist, erhalten wurde.
-
(8-2) Herstellung der
Zellinie, die I5E stabil exprimieren kann
-
Menschentypische
I5E-Rezeptor-cDNA (SEQ ID NR. 35) wurde durch bekanntes PCR geklont,
und in pAKKO1.1H-Expressionsvektor eingeführt. Der Expressionsvektor
wurde zu CHO/dhFr–-Zellen (American Type
Culture Collection) durch das in (3-5) oben beschriebenen Verfahren
transfektiert. Etwa 20 Kolonien der Transformanten-CHO-Zellen, die
10 bis 14 Tage nach der Initiierung der Inkubation wuchsen, wurden
ausgewählt.
Die ausgewählten
Transformanten wurden in eine 96-Lochplatte mit 3 × 104 Zellen/Loch inokuliert, und die Reaktivität mit MIT1
wurde gemäß dem in
(3-5) beschriebenen Testverfahren untersucht. Der I5E-exprimierte
CHO-Zellen-Klon Nr. 4 (bezeichnet als I5E-4-Zelle) mit einer guten
Reaktivität
wurden gescreent. Die Aminosäuresequenz,
die durch die Basensequenz kodiert wird, die durch SEQ ID NR. 35
dargestellt ist, wird durch SEQ ID NR. 36 gezeigt.
-
(8-3) Assay für die den
ZAQ-Rezeptor und den I5E-Rezeptor aktivierende Funktion von menschentypischen ZAQ-Ligandenpeptid
und MIT1
-
Das
menschentypische ZAQ-Ligandenpeptid, gereinigt durch das in BEISPIEL
7 beschriebene Verfahren, und das Schlangengift MIT1, gereinigt
durch das in (8-1) oben beschriebene Verfahren, wurden hinsichtlich
der ZAQ-Rezeptor-aktivierenden Funktion und der I5E-Rezeptor-aktivierenden
Funktion durch die in (3-5) beschriebenen Testverfahren analysiert.
Die Ergebnisse werden in 12 und 13 gezeigt.
-
Infolgedessen
induzierten das menschentypische ZAQ-Ligandenpeptid und Schlangengift
MIT1 konzentrationsabhängig
eine Erhöhung
der intrazellulären
Calciumionenkonzentration. Die ZAQ-Rezeptor-aktivierende Funktion
von Schlangengift MIT1 war zehnmal wirksamer als die von menschentypischem
ZAQ-Ligandenpeptid. Ebenso war die menschentypische I5E-Rezeptor-aktivierende
Funktion von Schlangengift MIT1 etwa 100 Mal wirksamer als die von
menschentypischem ZAQ-Ligandenpeptid.
-
BEISPIEL 9
-
Klonen der cDNA die das
Rattengehirn-abgeleitete neue G-Protein-gekoppelten Rezeptorprotein
(rZAQ1) kodiert, und Bestimmung der Basensequenz
-
Unter
Verwendung der gesamten Rattengehirn-cDNA-Bibliothek (CLONTECH Laboratories,
Inc.) als eine Matrize und zwei Primern (SEQ ID NR. 37 und SEQ ID
NR. 38) wurde PCR durchgeführt.
Die Reaktionslösung
in der obigen Reaktion bestand aus der obigen cDNA, verwendet als
eine 1/10 Volumen Matrize, 1/50 Volumen von Advantage 2 cDNA Polymerase
Mix (CLONTECH Laboratories, Inc.), jeweils 0,2 μM der jeweiligen Primer, 200 μM dNTPs und
einen Puffer, zugeführt
mit dem Enzym, um das Endvolumen von 25 μl zu erreichen. Bei der PCR
wurde (1) nach dem Umsetzen bei 94 °C für 2 Minuten (2) eine Zyklusgruppe
bei 94 °C für 20 Sekunden,
gefolgt von 72 °C
für 1 Minute
und 30 Sekunden dreimal wiederholt, wurde (3) eine Zyklusgruppe
bei 94 °C
für 20
Sekunden und bei 68 °C
für 1 Minute
und 30 Sekunden dreimal wiederholt, wurde (4) eine Zyklusgruppe
bei 94 °C
für 20
Sekunden, bei 62 °C
für 20
Sekunden und bei 68 °C
für 1 Minute
36 Mal wiederholt, und schließlich
wurde eine Extensionsreaktion bei 68 °C für 7 Minuten durchgeführt. Nach
der Beendigung der PCR wurde das Reaktionsprodukt zu dem Plasmidvektor
pCR2.1-TOPO (Invitrogen, Inc.) gemäß den Instruktionen, die dem
TA-Klonierungskit beiliegen (Invitrogen, Inc.), subkloniert, der
dann in Escherichia coli DH5α eingeführt wurde,
und die Klone, die die cDNA trugen, wurden in LB-Agarmedium, das
Ampicillin enthielt, selektiert. Die Sequenz von jedem Klon wurde
analysiert, wodurch cDNA erhalten wurde, die das neue G-Protein-gekoppelte
Rezeptorprotein (SEQ ID NR. 39) kodiert. In der Aminosäuresequenz,
abgeleitet von der Basensequenz dieser cDNA, wurde eine Homologie
von 83,7 % zu der Aminosäuresequenz,
die durch SEQ ID NR. 1 dargestellt ist, beobachtet. Das neue G-Proteingekoppelte
Rezeptorprotein, enthaltend diese Aminosäuresequenz, wurde rZAQ1 genannt.
Die Transformante (E. coli), die die DNA trägt, die die Basensequenz enthält, welche
durch SEQ ID NR. 39 dargestellt ist, wurde Escherichia coli DH5α/pCR2.l-rZAQ1
genannt.
-
BEISPIEL 10
-
Klonen der cDNA, die Rattengehirn-abgeleitetes
neues G-Protein-gekoppeltes Rezeptorprotein (rZAQ2) kodiert, und
Bestimmung der Basensequenz
-
Der
Klon, der rZAQ2 kodiert, wurde durch das Gen-Trapper-Verfahren erhalten.
Das heißt,
Sonden (SEQ ID NR. 41 und SEQ ID NR. 42) wurden biotiniert, und
zur einsträngigen
Gesamt-Rattenhirn-cDNA-Bibliothek (GIBCO-BRL) hybridisiert. Das
erhaltene einsträngige
Gen wurde zum Doppelstrang wiederhergestellt. Dieses Gen wurde zu
Escherichia coli DH10B elektroporiert, und Transformanten wurden
unter Verwendung von Ampicillinresistenz als ein Indikator erhalten.
Gemäß dem Kolonie-PCR
unter Verwendung einer Sonde (SEQ ID NR. 41) und einem Primer (SEQ
ID NR. 45) wurde ein Klon, der die Zielbasensequenz kodiert, selektiert.
Die Aminosäuresequenz
(SEQ ID NR. 47), abgeleitet von der Basensequenz (SEQ ID NR. 46)
von ORF (offenes Leseraster), vorhergesehen aus der Basensequenz
dieses Klons, wies eine Homologie von 80,6 % zu rZAQ1 auf. Das neue
G-Proteingekoppelte Rezeptorprotein, enthaltend diese Aminosäuresequenz,
wurde rZAQ2 genannt. Die Transformante (E. coli), die durch das
Gen-Trapper-V erfahren erhalten wurde, wurde Escherichia coli DH10B/pCMV-rZAQ2
genannt.
-
BEISPIEL 11
-
Kontraktionstest unter
Verwendung eines Probenpräparats
des Krummdarms, extrahiert aus dem Meerschweinchen
-
Meerschweinchen
(Std: Hartley, 7–8
Wochen alt, männlich,
wiegen etwa 450 g) verbluteten mittels Durchschneiden der Halsschlagader
mit Scheren, und der Bauch wurde geöffnet, um den Krummdarm zu
entfernen. Der Krummdarm wurde in eine Glaspetrischale gelegt, mit
Tyrode-Lösung
versetzt (Zusammensetzung: 138 mM NaCl, 2,7 mM KCl, 1,8 mM CaCl2, 0,5 mM MgCl2,
1,1 mM NaH2PO4,
11,9 mM NaHCO3, 5,6 mM Glukose), und Fettgewebe
und Bindegewebe, die am Krummdarm hafteten, wurden mit chirurgischen
Scheren und Pinzetten beschnitten. Der Krummdarm wurde dann in Stücke mit
etwa 1,5 cm Länge
geschnitten und als ein Probenpräparat
bereitgestellt. Dieses Präparat
wurde in einem Organbad (20 ml) suspendiert, das mit der Tyrode-Lösung, erwärmt auf
37 °C, gefüllt war,
und eine Last von 0,5 g wurde zur Stabilisierung über 30 Minuten
oder mehr aufgebracht.
-
Eine
Kontraktionsreaktion des Krummdarmpräparats wurde unter Verwendung
von AMPLIFIER CASE 7747, hergestellt von NEC San-Ei Co., Ltd., gemessen.
-
Nachdem
1 μM Acetylcholin
zugegeben wurde, um die maximale Kontraktionsreaktion zu bestimmen, wurde
das menschentypische ZAQ-Ligandenpeptid (ZAQL-1), erhalten gemäß der Verfahrensweise
von REFERENZBEISPIEL 1, oder Schlangengift MIT1, erhalten in (8-1-3)
oben, kumulativ verabreicht, um die Kontraktionsreaktion zu analysieren.
Zur Verdünnung
von ZAQL-1 und MIT1 wurde physiologische Kochsalzlösung, die
0,05 % Rinderserumalbumin enthält,
verwendet.
-
Die
Ergebnisse werden in 14 gezeigt. Die Kontraktionsreaktion,
induziert durch ZAQL-1 und MIT1, wird in Prozent dargestellt, wenn
die Kontraktionsreaktion, induziert durch 1 μM Acetylcholin, 100 % ausmachte.
-
Im
Ergebnis induzierten ZAQL-1 und MIT1 eine wirksame dosisabhängige Kontraktionsreaktion.
Die EC50-Werte von ZAQL-1 und MIT1, berechnet
aus der Dosis-Wirkungs-Kurve, betrugen 1,79 nM bzw. 0,40 nM.
-
BEISPIEL 12
-
Bindingsassay unter Verwendung
der ZAQ- und I5E-exprimierten CHO-Zellmembran-Fraktionen
-
(12-1) Herstellung von 125I-MIT1
-
Benzol,
enthaltend in [125I]-Bolton-Hunter Reagent
(monoiodiert, 37 MBq, NEN Co., NEX 120), wurde durch Stickstoffgas
entfernt, 20 μl
DMSO wurden zugegeben, und die eingedampften Stoffe wurden durch
Pipettieren gelöst.
Zu der Lösung
wurden 80 μl
der Lösung,
enthaltend 4 nmol MIT1, verdünnt
mit Boratpuffer (Zusammensetzung: 100 mM H3BO3, pH 8,5), zugegeben, und direkt danach
wurden sie gemischt, gefolgt von Inkubation bei Raumtemperatur für 2 Stunden.
Danach wurden 200 μl
10 % Acetonitril-0,1 % Trifluoressigsäure zugegeben und das Gemisch
wurde als eine Probe für
die HPLC-Fraktionierung bereitgestellt. Die Probe wurde auf eine
Säule (Toso,
Co., Ltd., 0,46 cm × 10
cm) zur TSKgel-Super-ODS-Umkehrphasen-Hochleistungsflüssigchromatographie
geladen, die mittels Führen
von 100 Vol.-% Elutionsmittel A (10 % Acetonitril/0,1 % Trifluoressigsäure)/0 Vol.-%
Elutionsmittel B (0,1 % Trifluoressigsäure/40 % Acetonitril) bei Raumtemperatur bei
einer Fließgeschwindigkeit
von 1 ml/min äquilibriert
wurde. Danach wurde die Konzentration an Elutionsmittel B auf 60
Vol.-% Elutionsmittel A/40 Vol.-% Elutionsmittel B über 1 Minute
erhöht
und dann mit einem linearen Gradienten auf 40 Vol.-% Elutionsmittel
A/60 Vol.-% Elutionsmittel B über
60 Minuten bei einer Fließgeschwindigkeit
von 1 ml/min erhöht.
Das eluierte [125I]-Bolton-Hunter Reagent-eingeführte MIT1
wurde manuell fraktioniert und mit einem Puffer verdünnt (Zusammensetzung:
20 mM Tris, 1 mM EDTA, 0,2 % BSA, 0,1 % CHAPS, 0,03 % NaN3, pH 7,4). Die Verdünnung wurde dispensiert und
bei –80 °C gelagert.
-
(12-2) Herstellung von
Zellmembranfraktionen
-
Zellmembranfraktionen
wurden gemäß dem Verfahren,
das in Journal of Pharmacology and Experimental Therapeutics, 279,
675–685,
1996 beschrieben ist, hergestellt. In Dulbecco's Modifiziertem Eagle-Medium (DMEM)
(Nissui Pharmaceutical Co., Ltd.), ergänzt mit 10 % dialysiertem fetalem
Rinderserum (JRH BIOSCIENCES), 1 mM MEM nichtessentieller Aminosäurelösung, 100
Einheiten/ml Penicillin und 100 μg/ml Streptomyzin,
wurden die Rezeptor-exprimierten CHO-Zellen, nämlich ZAQC-B1-Zellen oder I5E-4-Zellen,
in Single Trays (Nunc) kultiviert. Wenn die Zellen eine Zelldichte
von 80–90
% erreichten, wurde der Kulturüberstand
verworfen, und 30 ml PBS (TaKaRa Shuzo Co., Ltd.), enthaltend 1
g/l EDTA, wurden zu der Schale („Tray") zugegeben, um die Zellen zu waschen.
Dann wurde der Kulturüberstand
verworfen, und 30 ml des oben beschriebenen PBS-EDTA wurden zugegeben,
wobei er bei Raumtemperatur stehengelassen wurde. Die Schale wurde
geschüttelt,
um die Zellen abzulösen,
und die Zellsuspension wurde gewonnen. Erneut wurden 10 ml des oben
beschriebenen PBS-EDTA zu der Schale zum Waschen zμgμgegeben und
die Zellen, die auf der Schale verblieben, wurden abgelöst und mit
den Zellen in der zuvor gewonnen Zellsuspension gemischt. Die gemischte
Zellsuspension wurde bei 1.000 U/min für 5 Minuten unter Verwendung
einer Hochgeschwindigkeitszentrifuge (TOMY RL601) zentrifugiert.
Der Kulturüberstand
wurde verworfen, und die ausgefällten
Zellen wurden bei –80 °C gelagert.
Nachdem 4 ml eines Homogenatpuffers (Zusammensetzung: 10 mM NaHCO3, 5 mM EDTA, 0,5 mM Phenylmethylsulfonylfluorid
(PMSF), 10 μg/ml
Pepstatin-A, 20 μg/ml
Leupeptin, 10 μg/ml
E-64) zu den Niederschlägen
zugegeben wurden (entsprechend einer Single Tray), wurden die Niederschläge durch
Pipettieren suspendiert und die Suspension wurde unter Bedingungen
bei Bewertungspunkt 4 für
20 Sekunden unter Verwendung von Polytron (Schaufeltyp PTA7) homogenisiert,
was dreimal wiederholt wurde. Dann wurde das Homogenat bei 2.500
U/min für
10 Minuten unter Verwendung von Nr. 26 Rotor von Hitachi Hochgeschwindigkeitskühlzentrifuge
CR26H zentrifugiert. Der erhaltene Überstand wurde bei 35.000 U/min
für eine
Stunde unter Verwendung von SRP70AT Rotor der Ultrazentrifuge (Hitachi
Koki, SCP70H) zentrifugiert. Zu den erhaltenen Niederschlägen wurden
5 ml eines Bindungsassaypuffers zugegeben (Zusammensetzung: 20 mM
Tris, 1 mM EDTA, 0,5 mM PMSF, 10 μg/ml
Pepstatin-A, 40 μg/ml
Leupeptin, 10 μg/ml E-64,
0,03 % NaN3, pH 7,4), um sie zu suspendieren.
Die Suspension wurde durch Cell Strainer (FALCON 2350) filtriert,
wodurch die ZAQC-B1-Zellmembranfraktion (d. h. die ZAQ-Membranfraktion)
oder I5E-4-Zellmembranfraktion
(d. h. die I5E-Membranfraktion) erhalten wurde. Jede der Membranfraktionen
wurde hinsichtlich ihres Proteinniveaus unter Verwendung von Coomassie-Proteinassayreagens
(PIERCE) analysiert, und jeweils 100 μl wurden dispensiert und bei –80 °C gelagert.
-
(12-3) Bindungsassay
-
Die
ZAQ-Membranfraktion und die I5E-Membranfraktion wurden mit 0,1 %
BSA-enthaltendem
Bindungsassaypuffer, der in (12-2) oben beschrieben wurde, auf 10 μg/ml bzw.
20 μg/l
verdünnt,
und jeweils 200 μl
wurden in Röhrchen
dispensiert (FALCON 2053). Zu jeder Verdünnung der Membranfraktionen
wurden 2 μl einer
Testverbindung bzw. 2 μl
10 nM 125I-MIT1 zugegeben, gefolgt von Inkubation
bei 25 °C
für eine
Stunde. Als die Testverbindung wurde menschentypisches ZAQ-Ligandenpeptid
(ZAQL-1), erhalten gemäß der Verfahrensweise
von REFERENZBEISPIEL 1, oder Schlangengift MIT1 (nicht-markiertes
MIT1), erhalten in (8-1-3) oben, verwendet.
-
Dann
wurden 1,5 ml eines filtrierenden Puffers (Zusammensetzung: 20 mM
Tris, EDTA, 0,1 % BSA, 0,05 % CHAPS, 0,03 % NaN3,
pH 7,4) zur der Reaktionslösung
zugegeben. Das Gemisch wurde direkt durch ein Glasfaserfilterpapier
GF/F (Whatman) filtriert, der zuvor mit einem Puffer behandelt wurde
(Zusammensetzung: 20 mM Tris, 0,3 % Polyethylenimin, pH 7,4), 1,5
ml des filtrierenden Puffers wurden erneut zu den Reaktionsröhrchen zugegeben,
und das Gemisch wurde ebenso filtriert. Die Radioaktivität auf dem
Glasfaserfilterpapier wurde unter Verwendung eines γ-Zählers gemessen
(COBRA, Packard), wodurch die Bindungsmenge von 125I-MIT1
bestimmt wurde. Die 125I-MIT1-Bindungsmenge,
wenn nicht-markiertes
MIT1 (Endkonzentration von 1 μM)
als eine Testverbindung verwendet wurde, wurde als nicht-spezifische
Bindungsmenge (NSB) verwendet, die 125I-MIT1-Bindungsmenge,
wenn keine Testverbindung verwendet wurde, wurde als Gesamtbindungsmenge
(TB) verwendet, und die maximale Bindungsmenge (TB – NSB) wurde
berechnet. Ebenso wurde in bezug auf die Bindungsmenge (B), die
erhalten wurde, wenn eine Testverbindung zugegeben wurde, die Bindungsmenge
in Gegenwart einer Testverbindung durch den Prozentsatz einer spezifischen
Bindungsmenge (B – NSB),
berechnet auf die gesamte spezifische Bindung {%SPB = (B – NSB)/(TB – NSB) × 100}, ausgedrückt.
-
Die
Ergebnisse, die unter Verwendung der ZAQ-Membranfraktion und der
I5E-Membranfraktion erhalten wurden, werden in 15 bzw. 16 gezeigt.
-
Die
IC50-Werte des nicht-markierten MIT1, berechnet
durch dieses Bindungsassaysystem, betrugen 38 pM (ZAQ-Membranfraktion)
und 32 pM (I5E-Membranfraktion). Die IC50-Werte von ZAQL-1
betrugen 35 pM (ZAQ-Membranfraktion) und 93 pM (I5E-Membranfraktion).
-
REFERENZBEISPIEL 1
-
Herstellung von menschentypischem
ZAQ-Ligand (SEQ ID NR. 21) in Escherichia coli
-
(REFERENZBEISPIEL 1-1)
-
Konstruktion
von menschentypischem ZAQ-Ligand-exprimierenden Plasmid in Escherichia
coli
-
(a) Unter Verwendung der
folgenden sechs DNA-Fragmente #1 bis #6 wurde die Struktur-DNA des ZAQ-Liganden
hergestellt.
-
-
(b) Phosphorylierung von
DNA-Oligomer
-
Jeder
der vier DNA-Oligomere (#2 bis #5), außer #1 und #6, die das 5'-Ende bilden sollten,
wurde bei 37 °C
für eine
Stunde in 25 μl
Reaktionslösung
zur Phosphorylierung umgesetzt [10 μg DNA-Oligomer, 50 mM Tris-HCl,
pH 7,6, 10 mM MgCl2, 1 mM Sperimidin, 10
mM Dithiothreitol (hierin nachstehend als DTT abgekürzt), 0,1
mg/ml Rinderserumalbumin (hierin nachstehend als BSA abgekürzt), 1
mM ATP, 10 Einheiten von T4-Polynukleotidkinase (TaKaRa Shuzo Co.,
Ltd.)], um das 5'-Ende
von jedem Oligomer zu phosphorylieren. Nach der Phenolbehandlung,
gefolgt von der Zugabe vom 2fachen Volumen an Ethanol wurde das
Gemisch auf –70 °C abgekühlt und
zentrifugiert, wodurch DNA ausfiel.
-
(c) Ligation des DNA-Fragments
-
Das
oben in (b) erhaltene DNA-Fragment wurde mit #1 und #6 oben kombiniert,
um das Volumen von 120 μl
zu erhalten. Das Gemisch wurde zum Annealing bei 90 °C für 10 Minuten
gehalten und allmählich
auf Raumtemperatur abgekühlt.
Unter Verwendung von TaKaRa DNA Ligation Kit ver. 2 (TaKaRa Shuzo
Co., Ltd.) wurde die Ligation durchgeführt. Nachdem 30 μl der Lösung II,
zugeführt
mit dem Kit, zu 30 μl
der Annealing-Lösung
zugegeben wurden, um es gründlich
zu mischen, wurden 60 μl
der Lösung
I, zugeführt
mit dem Kit, zu dem Gemisch zugegeben, und das Gemisch wurde bei
37 °C für eine Stunde
zur Ligation umgesetzt, gefolgt von Phenolbehandlung. Die wässerige
Phase wurde gewonnen und 2 Volumen an Ethanol wurden dazugegeben.
Das Gemisch wurde auf –70 °C abgekühlt und
dann zentrifugiert, wodurch die DNA ausfiel. Das so erhaltene DNA-Fragment
wurde unter Verwendung von T4-Polynukleotidkinase phosphoryliert
(TaKaRa Shuzo Co., Ltd.), und dann für den folgenden Schritt (d)
bereitgestellt.
-
(d) Konstruktion des ZAQ-Ligandenexpressionsvektors
-
Nachdem
pTCII (beschrieben in der japanischen offengelegten Patentanmeldung
Nr. 2000-178297) mit
NdeI und BamHI (TaKaRa Shuzo Co., Ltd.) bei 37 °C für 2 Stunden digeriert wurde,
wurde das DNA-Fragment von 4,3 kb, das durch 1 % Agarosegelelektrophorese
getrennt wurde, unter Verwendung von QIAquick Gel Extraction Kit
(QIAGEN) extrahiert und in 25 μl
TE-Puffer gelöst.
Die NdeI- und BamHI-Fraktionen von diesem pTCII wurden mit dem Strukturgen
(SEQ ID NR. 58) vom oben hergestellten ZAQ-Liganden unter Verwendung
von TaKaRa DNA Ligation Kit ver. 2 (TaKaRa Shuzo Co., Ltd.) ligiert,
wodurch der Expressionsvektor hergestellt wurde. Unter Verwendung
von 10 μl
der Reaktionslösung
wurden E. coli JM109 kompetente Zellen (TOYOBO) transformiert und
auf LB-Agarmedium, enthaltend 10 μg/ml
Tetracyclin, inokuliert, gefolgt von Inkubation bei 37 °C über Nacht.
Die gebildete Tetracyclin-resistente Kolonie wurde ausgewählt. Die
Transformante wurde in LB-Medium über Nacht
inkubiert, und Plasmid pTCh1ZAQ wurde unter Verwendung von QIAprep8
Miniprep Kit (QIAGEN) hergestellt. Die Basensequenz von dieser ZAQ-Liganden-DNA wurde unter Verwendung
von Model 377 DNA Sequenzer von Applied Biosystems, Inc. identifiziert.
Plasmid pTCh1ZAQ wurde zu Escherichia coli MM294 (DE3) transfektiert,
um den ZAQ-Ligandexpressionstamm, Escherichia coli MM294 (DE3)/pTCh1ZAQ,
zu erhalten.
-
(REFERENZBEISPIEL 1-2)
-
Herstellung des ZAQ-Liganden
-
In
einem 2-Liter-Kolben wurde oben beschriebenes Escherichia coli MM294
(DE3)/pTCh1ZAQ unter Schütteln
in 1 Liter des LB-Mediums (1 % Pepton, 0,5 % Hefeextrakt und 0,5
% Natriumchlorid), ergänzt
mit 5,0 mg/l Tetracyclin, bei 37 °C
für 8 Stunden
kultiviert. Das erhaltene kultivierte Medium wurde zu einem Fermentationsbehälter mit
50 l Volumen übertragen,
der mit 19 l Hauptfermentationsmedium beschickt ist (1,68 % Natriummonohydrogenphosphat,
0,3 % Kaliumdihydrogenphosphat, 0,1 % Ammoniumchlorid, 0,05 % Natriumchlorid,
0,05 % Magnesiumsulfat, 0,02 % Antischaummittel, 0,00025 % Eisen(II)-sulfat,
0,0005 % Thiaminhydrochlorid, 1,5 % Glukose, 1,5 % Hy-Case Amino),
und das Rühren
an der Luft wurde bei 30 °C
initiiert. Wenn die Trübheit
des kultivierten Mediums 500 Klett erreichte, wurde Isopropyl-β-D-thiogalactopyranosid
in einer Endkonzentration von 12 mg/l zugegeben, und die Inkubation
wurde für
weitere 4 Stunden fortgesetzt. Nach der Beendigung der Kultur wurde
das Kulturmedium zentrifugiert, wodurch etwa 200 g nasse Zellen
erhalten wurden. Die Zellen wurden bei –80 °C gelagert.
-
Diese
Transformante Escherichia coli MM294 (DE3)/pTCh1ZAQ ist im Institute
for Fermentation, Osaka (IFO) unter der Zugangsnummer IFO 16527
hinterlegt.
-
(REFERENZBEISPIEL 1-3)
-
Aktivierung des ZAQ-Liganden
-
Mittels
Zugeben von 400 ml eines Gemisches aus 200 mM Tris/HCl und 7 M Guanidinhydrochlorid
(pH 8,0) zu 200 g der in REFERENZBEISPIEL (1-2) erhaltenen Zellen
wurden die Zellen darin gelöst,
und die Lösung
wurde zentrifugiert (10.000 U/min, 1 Stunde). Zu dem Überstand
wurden 10 l eines Gemisches aus 0,4 M Arginin, 50 mM Tris/HCl, 0,2
mM GSSG und 1 mM GSH (pH 8,0) zugegeben, und das Gemisch wurde bei 4 °C über Nacht
zur Aktivierung gehalten.
-
(REFERENZBEISPIEL 1-4)
-
Reinigung des ZAQ-Liganden
-
Die
regenerierende Lösung
nach der Aktivierung in REFERENZBEISPIEL (1-3) wurde auf pH 6,0
eingestellt und auf einer SP-Sepharose-Säule (11,3 cm × 15 cm), äquilibriert
mit 50 mM Phosphatpuffer (pH 6,0), adsorbiert. Die Säule wurde
mit 600 mM NaCl/50 mM Phosphatpuffer (pH 6,0) eluiert, und die Fraktionen,
enthaltend den ZAQ-Liganden, wurden zusammengefaßt. Die Fraktion wurde durch
CM-5PW (21,5 mm × 150
mm Länge), äquilibriert
mit 50 mM Phosphatpuffer (pH 6,0), geführt und darauf adsorbiert.
Nach dem Waschen wurde die Elution mit einem schrittweisen Gradienten
(60 Minuten) von 0 bis 100 % B (B = 50 mM Phosphatpuffer + 1 M NaCl,
pH 6,05) durchgeführt,
wodurch die ZAQ-Ligandenfraktion
(Elutionszeit: ca. 40 Minuten) erhalten wurde. Diese Fraktion wurde
weiter durch C4P-50 (21,5 mm Innendurchmesser × 300 mm Länge, Showa Denko), äquilibriert
mit 0,1 % Trifluoressigsäure,
geführt
und darauf adsorbiert. Nach dem Waschen wurde die Elution mit einem
schrittweisen Gradienten (60 Minuten) von 25 bis 50 % B (B = 80
% Acetonitril/0,1 % Trifluoressigsäure) durchgeführt. Die
ZAQ-Ligandenfraktionen (Elutionszeit: ca. 40 Minuten) wurden zusammengefaßt und lyophilisiert,
wodurch 80 mg des lyophiliserten ZAQ-Ligandenpulvers erhalten wurden.
-
(REFERENZBEISPIEL 1-5)
-
Charakterisierung des
ZAQ-Liganden
-
(a) Analyse unter Verwendung
von SDS-Polyacrylamidgelelektrophorese
-
Der
ZAQ-Ligand, erhalten in dem REFERENZBEISPIEL (1-4), wurde in Probenpuffer
[Laemmli, Nature, 227, 680 (1979)], ergänzt mit 100 mM DTT, suspendiert.
Die Suspension wurde bei 95 °C
für 1 Minute erhitzt,
gefolgt von Elektrophorese auf Multigel 15/25 (Daiichi Kagaku Yakuhin).
Nach der Elektrophorese wurde das Gel mit Coomassie-Brilliantblau
eingefärbt,
und eine einzelne Proteinbande wurde an derselben Stelle als die
des COS7-Zellenabgeleiteten rekombinanten ZAQ-Ligandenpräparats,
erhalten in BEISPIEL 5, nachgewiesen. Basierend darauf wurde offenbart,
daß das
E. coli-abgeleitete rekombinante ZAQ-Ligandenpräparat, erhalten in REFERENZBEISPIEL
(1-4), eine sehr hohe Reinheit aufwies und dasselbe Molekulargewicht wie
das des rekombinanten ZAQ-Liganden, hergestellt aus COS7-Zellen,
aufwies.
-
(b) Analyse der Aminosäurezusammensetzung
-
Die
Aminosäurezusammensetzung
wurde unter Verwendung eines Aminosäureanalysators bestimmt (Hitachi
L-8500A Amino Acid Analyzer). Die Ergebnisse offenbaren, daß die Aminosäurezusammensetzung mit
der Aminosäurezusammensetzung übereinstimmte,
die aus der Basensequenz der DNA von ZAQ-Ligand abgeleitet wurde
(Peptid, umfassend die Aminosäuresequenz,
die durch SEQ ID NR. 21 dargestellt ist) (Tabelle 2). TABELLE
2
Säurehydrolyse
(Mittelwert in Hydrolyse mit 6N HCl-1 % Phenol bei 110 °C für 24 und
48 Stunden)
- 1)Wert, extrapoliert
bei 0 Stunden
- 2)nicht nachgewiesen
-
(c) Analyse der N-terminalen
Aminosäuresequenz
-
Die
N-terminale Aminosäuresequenz
wurde unter Verwendung eines Gasphasen-Proteinsequenzers bestimmt
(PE Applied Biosystems, Model 492). Im Ergebnis stimmte die Aminosäuresequenz
mit der N-terminalen Aminosäuresequenz
des ZAQ-Liganden überein,
der von der Basensequenz der erhaltenen ZAQ-Liganden-DNA abgeleitet
wurde (Tabelle 3). TABELLE
3
- 1)Analyse wurde
unter Verwendung von 150 pmol Phenylthiohydantoin durchgeführt. N.D.
nicht nachgewiesen
-
(d) Analyse der C-terminalen
Aminosäure
-
Die
C-terminale Aminosäuresequenz
wurde unter Verwendung einer Aminosäure bestimmt (Hitachi L-8500A
Amino Acid Analyzer). Der erhaltene ZAQ-Ligand stimmte mit der C-terminalen
Aminosäuresequenz überein,
die aus der Basensequenz von DNA abgeleitet ist (Tabelle 4). TABELLE
4
- Gasphasenhydrazinolyseverfahren (100 °C, 3,5 Stunden)
-
(e) Massenspektrometrie
-
Massenspektrometrie
wurde unter Verwendung von LCQ-Ion-Trap-Massenspektrometer (hergestellt von
ThermoQuest, Inc.), ausgestattet mit Nano-ESI-Ionenquellen, durchgeführt. Im
Ergebnis wurde das Molekulargewicht von 9657,55 ± 0,89 gefunden und stimmte
gut mit dem berechneten Molekulargewicht (9657,3) des ZAQ-Liganden,
der durch SEQ ID NR. 21 dargestellt ist, überein, worin 10 Cys-Reste,
die in SEQ ID NR. 21 enthalten sind, 5 Paare von Disulfidbindungen
bildeten.
-
(REFERENZBEISPIEL 1-6)
-
Assay für ZAQ-Liganden
(Assay für
die die intrazelluläre
Ca-Ionenkonzentration erhöhende
Aktivität
unter Verwendung von FLIPR)
-
Das
gereinigte rekombinante ZAQ-Ligandenpräparat, abgeleitet von Escherichia
coli, das in REFERENZBEISPIEL (1-4) hergestellt wurde, wurde hinsichtlich
der Aktivität
(Assay für
die die intrazelluläre
Ca-Ionenkonzentration erhöhende
Aktivität
unter Verwendung von FLIPR) durch das Verfahren in BEISPIEL 5 analysiert.
Im Ergebnis wies es eine Aktivität
auf, äquivalent
zu der des COS7-abgeleiteten rekombinanten Präparats (gereinigter ZAQ-Ligand), erhalten
in BEISPIEL 5.
-
INDUSTRIELLE
ANWENDBARKEIT
-
Das
Peptid der Erfindung, die DNA, die das Peptid der Erfindung kodiert
(hierin nachstehend manchmal nur als die erfindungsgemäße DNA bezeichnet),
und der Antikörper
zu dem Peptid der Erfindung (hierin nachstehend manchmal nur als
der erfindungsgemäße Antikörper bezeichnet)
sind zum Implementieren (1) von Mitteln zur Vorbeugung/Behandlung
von verschiedenen Krankheiten, die mit dem Peptid der Erfindung verbunden
sind; (2) Screenen einer Verbindung oder seines Salzes, die die
Bindungseigenschaft zwischen dem Peptid der Erfindung und dem Protein
der Erfindung verändert;
(3) Quantifikation des Peptids der Erfindung oder seines Salzes;
(4) ein Mittel zur Gendiagnose; (5) Arzneimittel, die die Antisense-DNA umfassen; (6)
Arzneimittel, die den erfindungsgemäßen Antikörper umfassen; (7) Präparieren
von nicht-menschlichen Tieren, die die erfindungsgemäße DNA tragen;
(8) Arzneimitteldesign, basierend auf dem Vergleich zwischen Liganden
und Rezeptoren, die strukturell analog sind, usw. nützlich [SEQUENZPROTOKOLL]
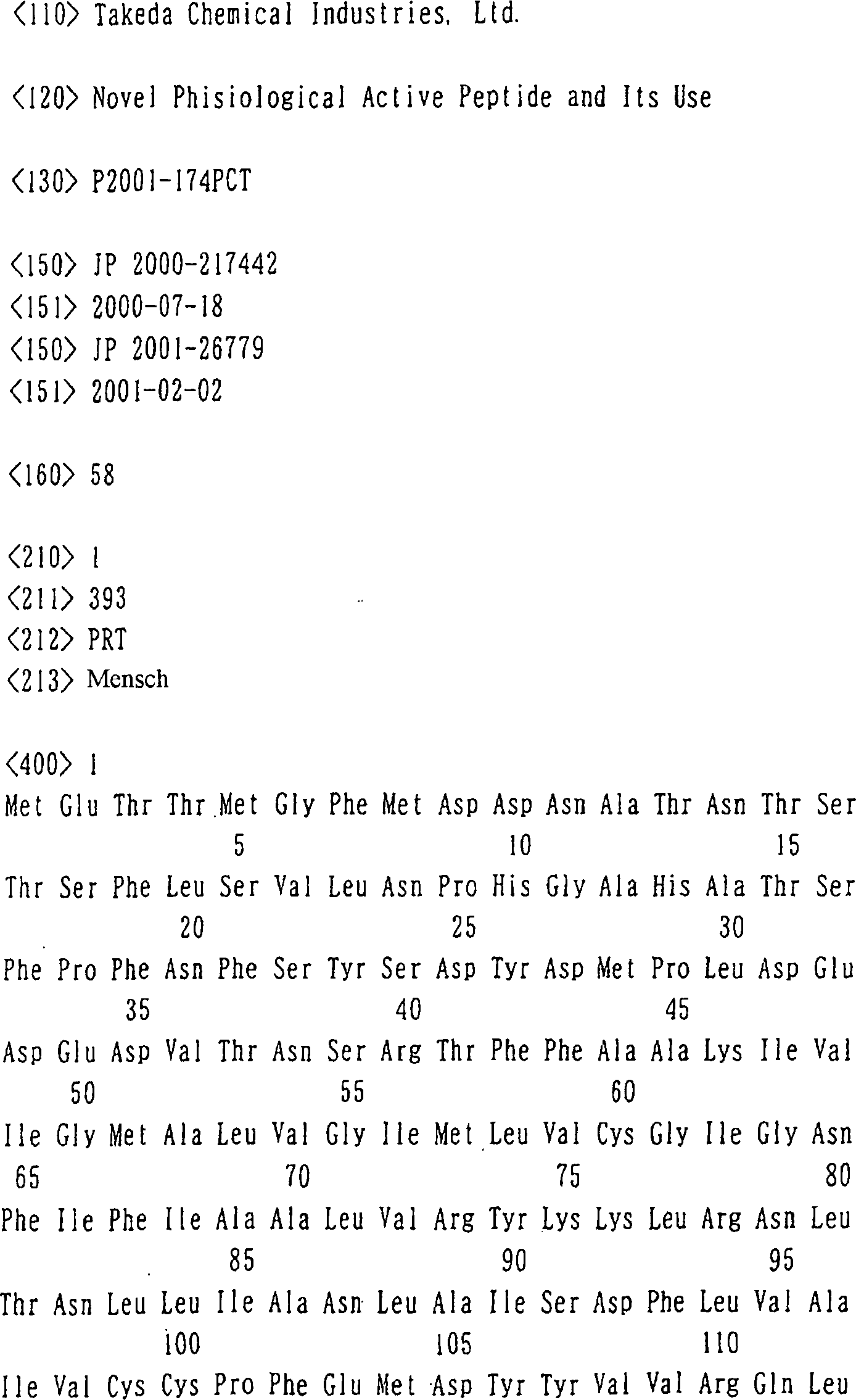
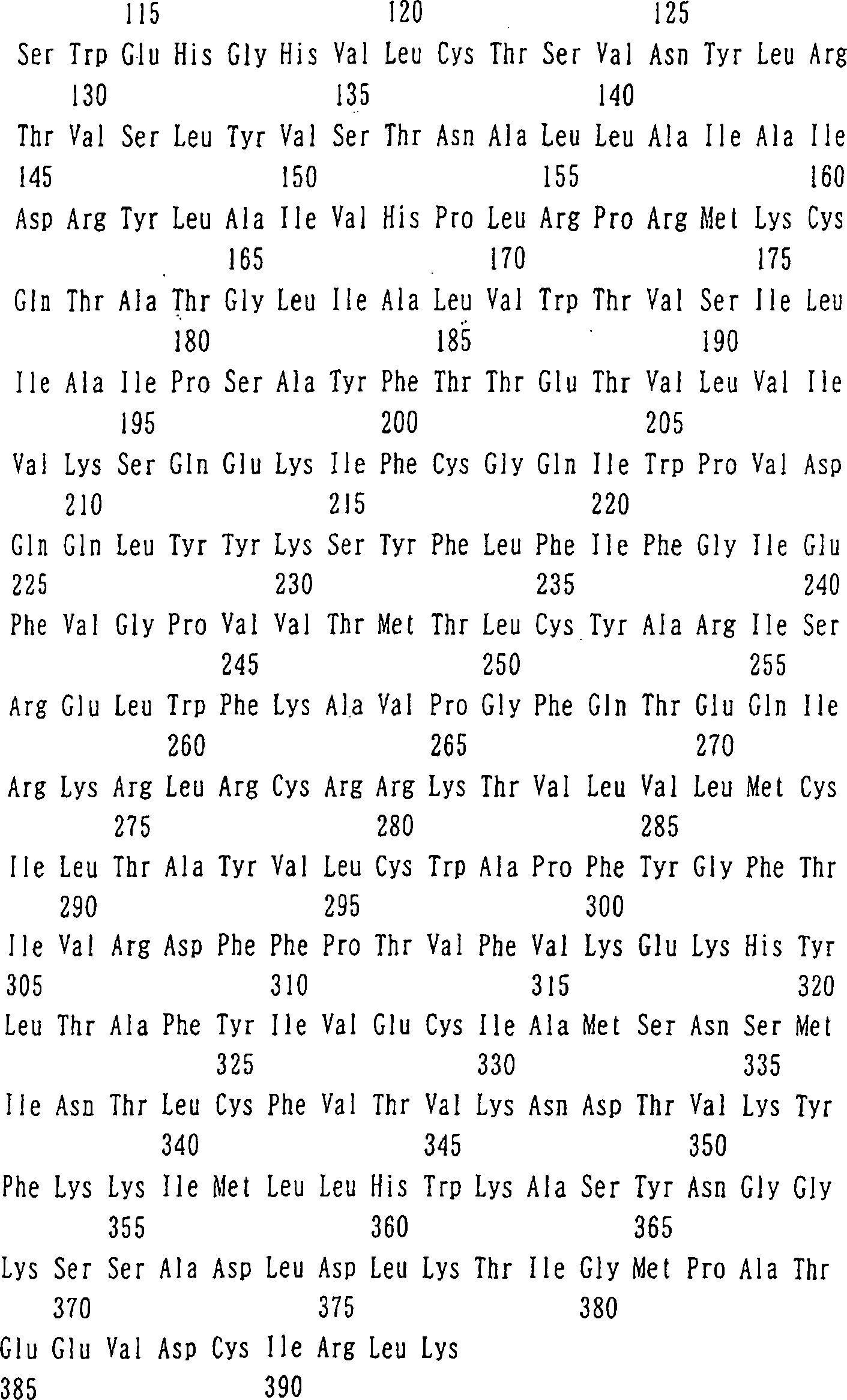













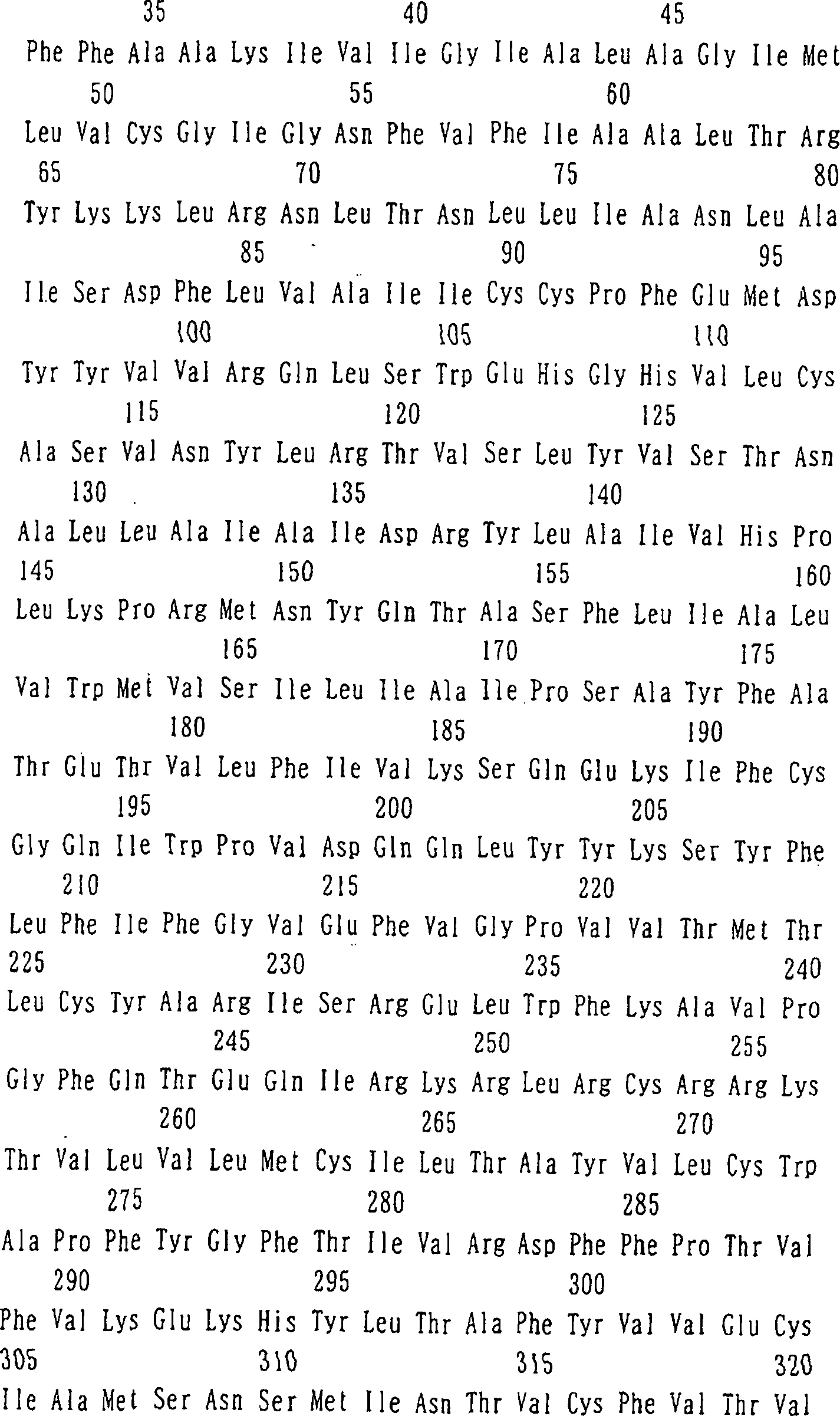






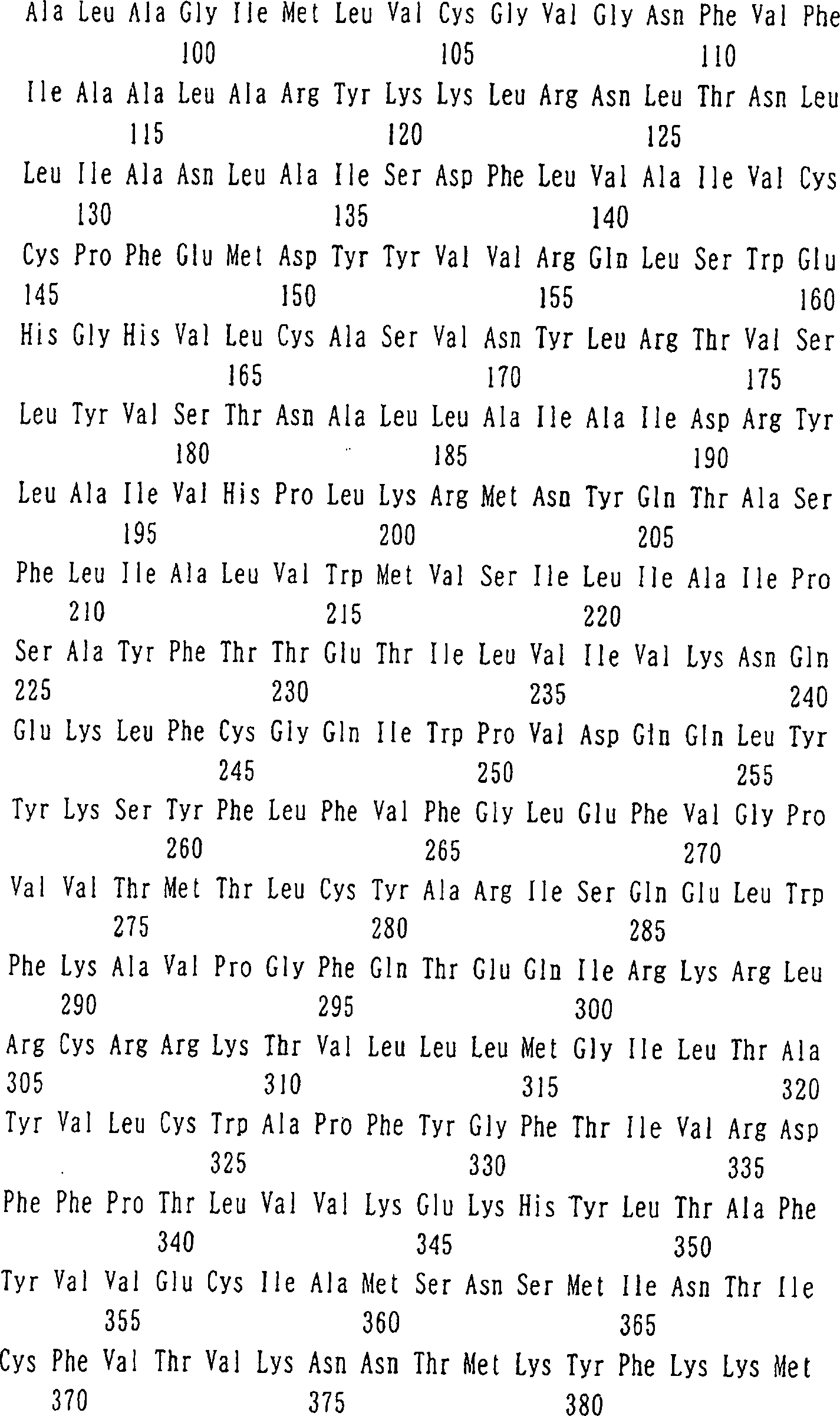
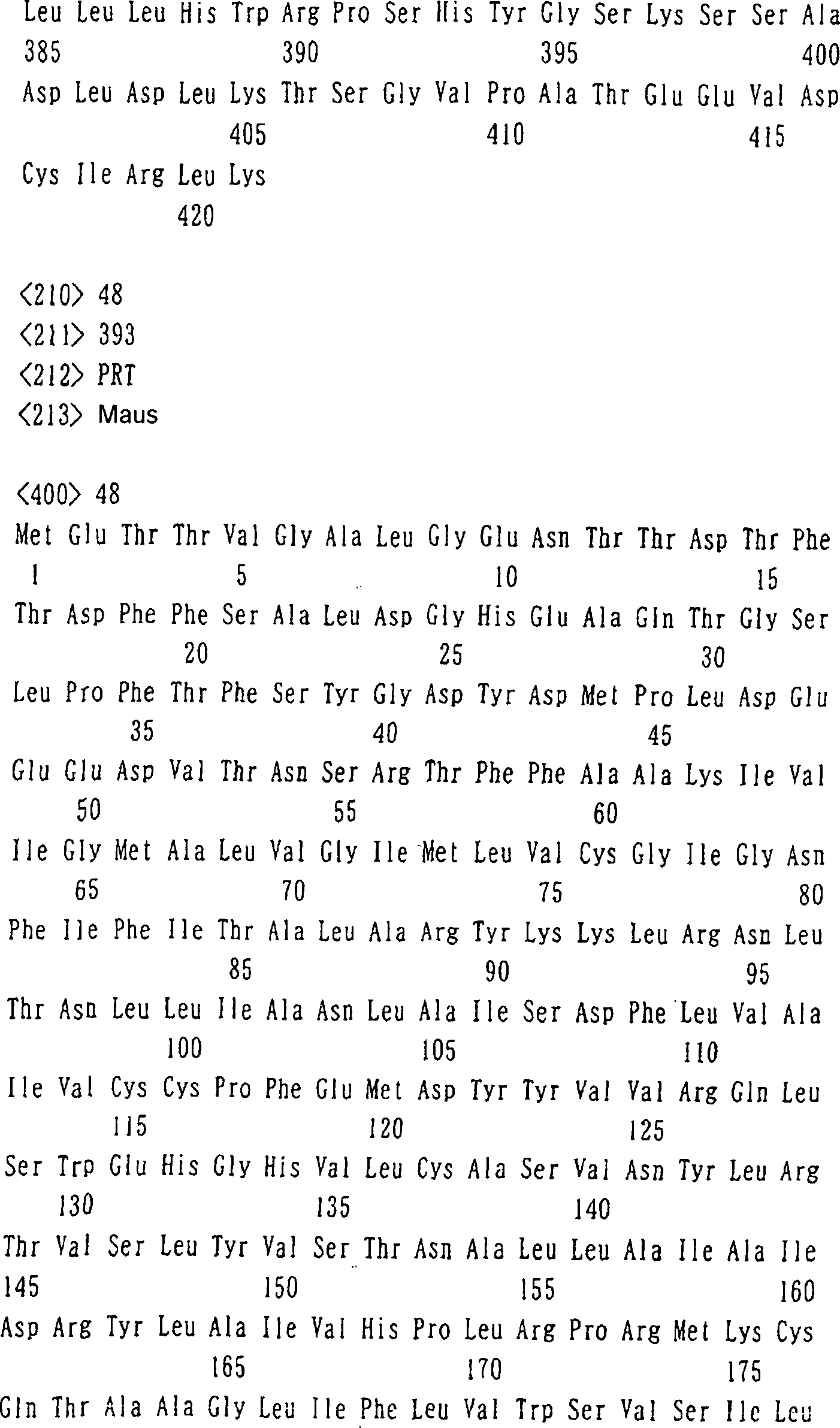

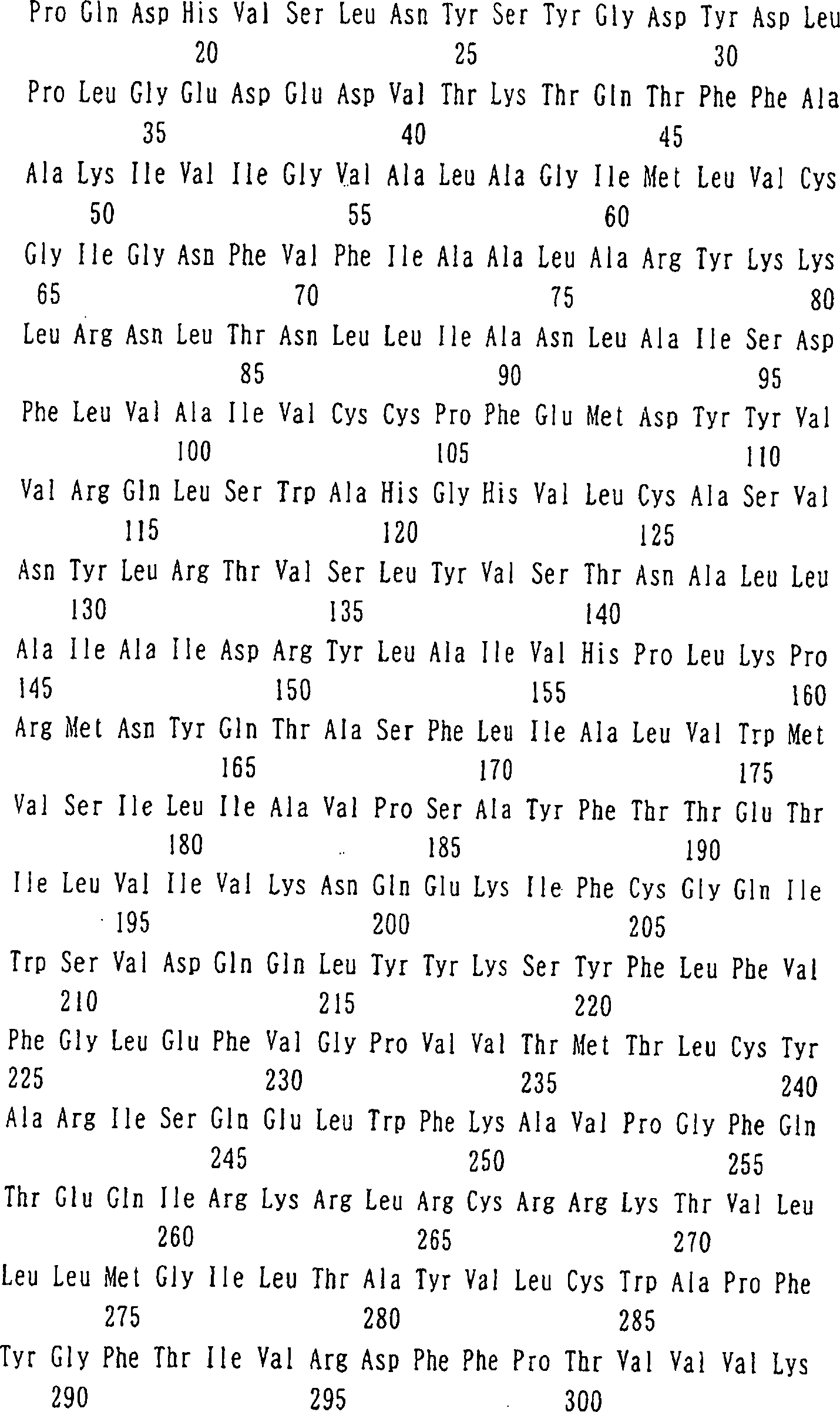





[SEQUENZPROTOKOLL]