CN103842379A - 用于生成单克隆抗体的方法和试剂 - Google Patents
用于生成单克隆抗体的方法和试剂 Download PDFInfo
- Publication number
- CN103842379A CN103842379A CN201280021326.1A CN201280021326A CN103842379A CN 103842379 A CN103842379 A CN 103842379A CN 201280021326 A CN201280021326 A CN 201280021326A CN 103842379 A CN103842379 A CN 103842379A
- Authority
- CN
- China
- Prior art keywords
- immunoglobulin
- antigen
- sequence
- chain
- variable region
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Pending
Links
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/08—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from viruses
- C07K16/081—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from viruses from DNA viruses
- C07K16/082—Hepadnaviridae, e.g. hepatitis B virus
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/44—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material not provided for elsewhere, e.g. haptens, metals, DNA, RNA, amino acids
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/20—Immunoglobulins specific features characterized by taxonomic origin
- C07K2317/21—Immunoglobulins specific features characterized by taxonomic origin from primates, e.g. man
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/56—Immunoglobulins specific features characterized by immunoglobulin fragments variable (Fv) region, i.e. VH and/or VL
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/56—Immunoglobulins specific features characterized by immunoglobulin fragments variable (Fv) region, i.e. VH and/or VL
- C07K2317/565—Complementarity determining region [CDR]
Abstract
在一个实施方案中,本发明涉及用于生成特异性结合抗原的单克隆抗体的方法。本发明可以从多克隆抗体群体诸如非特异性多克隆群体或特异性结合抗原的多克隆抗体群体开始。本方法包括获得编码来自动物的多种免疫球蛋白的重链和轻链免疫球蛋白链(或其可变区)的核酸分子;获得特异性结合所选抗原的多克隆免疫球蛋白群体的肽片段的质谱信息;将多克隆免疫球蛋白的肽片段的质谱信息与核酸序列编码的预测氨基酸序列的预测质谱信息相比较和/或关联,并且然后装配重链和轻链以产生特异性结合抗原的抗体(或其可变区)。
Description
交叉引用相关申请
本申请主张2011年3月9日提交的美国临时申请号61/450,922、2011年11月15日提交的美国临时申请号61/560,006、2011年12月5日提交的美国临时申请号61/566,876和2012年2月3日提交的美国临时申请号61/594,729的优先权,其中每个申请的完整内容通过引用并入本文。
发明领域
本公开涉及生物学,更具体地,涉及分子生物学和免疫学。
发明背景
抗体是生物学和商业上重要的多肽,其以高特异性和亲和力结合至被称为抗原的特定的目标分子。抗体由脊椎动物的免疫细胞产生,并且所有天然存在的抗体共有相同的基本结构,即两条相同的重链和与之共价结合的两条相同的轻链。单一重链和单一轻链的N-末端区域形成对于各单独抗体特有的抗原结合位点。重链的C-末端区域决定抗体的特定同种型,且相同的抗体产生细胞可以产生不同同种型的抗体,其中由细胞产生的所有抗体具有相同的抗原结合位点。不同同种型通常在动物中行使不同的功能。例如,E同种型的抗体(即IgE抗体)参与过敏性应答,而A同种型的抗体(即,IgA抗体)可以存在于粘膜、唾液和母乳中。四条链的抗体分子可以自身存在(例如IgG抗体),或与额外单体存在以形成二聚物(例如,IgA抗体)或者甚至五聚体(例如,IgM抗体)。
在很好理解抗体的基本结构的情况下,可以通过使用标准的分子生物学技术操作抗体的不同区域而产生重组抗体。例如,美国专利号6,180,370和6,548,640(完整地通过引用并入本文)描述了通过使用分子生物学技术操作非人抗体的各区域而使在非人动物中天然存在的抗体人源化。描述了使用标准的分子生物学技术用于操作或产生重组抗体的其他方法(参见,例如,PCT公开号WO91/17271、PCT公开号WO92/01047;美国专利号5,969,108、6,331,415、7,498,024、和7,485,291, 其中所有都完整地通过引用并入本文)。
在免疫应答过程中,动物将产生许多不同的抗体,每种具有不同的抗原结合特异性。抗体的该群体被称为抗体的多克隆群体。如果免疫应答针对特定抗原,则由动物产生的大多数(但不是所有)的多克隆抗体特异性结合该抗原。然而,由于对抗原的结合亲和力和结合位点上的差异,一些多克隆抗体比其他多克隆抗体更有利。在其1975年赢得诺贝尔奖的发现中,Kohler和Milstein发现了从产生多克隆抗体的动物分离和永生化单一抗体生成细胞的方法,所述单一抗体生成细胞产生特异性结合目标抗原的单克隆抗体(Kohler和Milstein, Nature 256: 495-497, 1975)。该永生化技术(其包括将抗体产生细胞与永生化细胞融合产生产生单克隆抗体的杂交瘤)在过去35年已经是制备单克隆抗体的行业标准。
尽管其具有广泛性和长久性,但是Kohler和Milstein杂交瘤方法具有许多缺点。例如,它非常费时费力。更贴切地,鉴于它是如何费时费力,只有一小部分动物的抗体产生细胞得到永生化且针对其产生特异性结合抗原的抗体的能力得到筛选。最后,哪怕只要一次分离具有所需抗原特异性的杂交瘤,但是获得抗体的氨基酸序列以促进抗体的进一步操作,诸如人源化,是费力费时的。
存在对于生成特异性结合所需抗原的单克隆抗体的改进方法的需要。
发明概述
本发明的各个方面和实施方案提供了迅速且准确生成特异性结合目标抗原的单克隆抗体的方法和系统。在进一步方面和实施方案中,本发明提供了用于进行本发明的各种方法的试剂和组合物,和由进行本发明的各种方法产生的试剂和组合物。在一些实施方案中,本文公开的方法、试剂和组合物可用于从受试者的循环中生成单克隆抗体。
在一个方面,本发明提供了用于获得特异性结合抗原的免疫球蛋白(或其可变区)的序列的方法,其包括:(a) 提供编码至少一种动物的多种免疫球蛋白的免疫球蛋白链(或其可变区)的核酸序列;(b) 获得特异性结合抗原的多克隆免疫球蛋白群体的免疫球蛋白重链和免疫球蛋白轻链的肽片段的质谱信息;(c) 将肽片段的质谱信息与核酸序列的预测质谱信息(所述预测质谱信息源自所述核酸序列的核苷酸序列编码的预测氨基酸序列)相关联,以鉴定编码包含所述肽片段的免疫球蛋白链(或其可变区)的核苷酸序列;和(d) 基于所述肽片段对所述免疫球蛋白链或其片段的氨基酸序列覆盖率从鉴定的免疫球蛋白链(或其可变区)的核苷酸序列或氨基酸序列中选择,以获得特异性结合抗原的免疫球蛋白的重或轻链的核苷酸序列或氨基酸序列。
在一些实施方案中,装配步骤(d)中选择的免疫球蛋白重链和免疫球蛋白轻链(或其可变区)以生成特异性结合抗原的免疫球蛋白(或其可变区)。
在一些实施方案中,在装配前,通过重组分子生物学技术或基因合成技术合成步骤(d)中获得的免疫球蛋白链可变区的核苷酸序列或氨基酸序列。
在一些实施方案中,所述方法进一步包括:用免疫测定筛选生成的免疫球蛋白(或其可变区),以确认所述免疫球蛋白(或其可变区)特异性结合抗原。在一些实施方案中,免疫测定选自流式细胞术测定、酶联免疫吸附测定(ELISA)、Western印迹测定、免疫组织化学测定、免疫荧光测定、放射免疫测定、中和测定、结合测定、亲和力测定、或蛋白或肽免疫沉淀测定。
在一些实施方案中,基于所述肽片段对一部分链(例如,可变区或互补决定区)的氨基酸序列覆盖率进行步骤(d)中的免疫球蛋白重链和免疫球蛋白轻链的选择。
在其他实施方案中,基于所述肽片段对免疫球蛋白链或其片段的氨基酸序列覆盖率组合至少一种参数进行步骤(d)中的免疫球蛋白重链或轻链的选择,所述参数选自绘制的独特肽数目、谱共享、总肽计数、独特肽计数、编码核酸序列的频率和克隆相关性。
在各个实施方案中,核酸序列和源自核酸序列的信息(包括,例如,核苷酸序列、预测的氨基酸序列、和预测的质谱)位于遗传物质数据库中。
在一些实施方案中,由其获得核酸序列的动物是暴露于抗原的动物。
在一些实施方案中,由编码来自动物的多种免疫球蛋白的免疫球蛋白链(或其可变区)的所述核酸序列编码的预测的氨基酸序列通过以下方式获得:(1) 从来自所述动物的白血细胞分离核酸分子;(2) 使用对于在所述编码免疫球蛋白链(或其可变区)的核酸分子邻近的多核苷酸序列特异性的引物扩增编码免疫球蛋白链(或其可变区)的核酸分子;(3) 获得编码来自所述动物的多种免疫球蛋白的免疫球蛋白链(或其可变区)的所述扩增的核酸分子的核苷酸序列;和(4) 使用遗传密码来将核苷酸序列翻译成预测的氨基酸序列。
在一些实施方案中,核酸序列是表达的核酸序列(例如,在动物细胞中转录成RNA和/或翻译成蛋白)。
在一些实施方案中,由编码来自动物的多种免疫球蛋白的免疫球蛋白链(或其可变区)的所述核酸分子编码的预测的氨基酸序列通过以下方式获得:(1) 从来自所述动物的白血细胞分离核酸分子; (2)使用对于在所述编码免疫球蛋白链(或其可变区)的核酸分子邻近的多核苷酸序列特异性的引物对编码免疫球蛋白链(或其可变区)的核酸分子进行测序,以获得编码来自所述动物的多种免疫球蛋白的免疫球蛋白链(或其可变区)的核苷酸序列; 和(3) 使用遗传密码来将核酸序列翻译成氨基酸序列。在一些实施方案中,核酸分子是RNA分子,且所述扩增步骤包括初始的逆转录步骤。
在一些实施方案中,与编码免疫球蛋白链(或其可变区)的核酸分子邻近的多核苷酸序列选自免疫球蛋白基因侧翼的基因组DNA、编码免疫球蛋白链恒定区的多核苷酸序列、和编码免疫球蛋白链构架区的多核苷酸序列。
在一些实施方案中,预测的质谱信息使用包括以下步骤的方法来获得:(i) 用一种或多种蛋白酶和/或一种或多种化学蛋白切割试剂进行核酸分子的核苷酸序列编码的预测氨基酸序列的理论消化来生成虚拟的肽片段;和 (ii) 生成所述虚拟肽片段的预测质谱。
在一些实施方案中,所述肽片段的观察的质谱信息使用包括以下步骤的方法来获得:(i) 分离特异性结合抗原的多克隆免疫球蛋白的群体;(ii) 用一种或多种蛋白酶和/或一种或多种化学蛋白切割试剂消化群体以生成片段;和(iii) 获得所述肽片段的质谱信息。在一些实施方案中,多克隆抗体的群体使用包括以下步骤的方法来分离:(1) 从动物获得体液或其部分(例如,血液、血清和/或血浆);(2) 在使特异性结合抗原的免疫球蛋白将变得与抗原结合的条件下使所述体液或其部分通过抗原;和(3) 收集与所述抗原结合的所述免疫球蛋白以获得特异性结合抗原的多克隆免疫球蛋白的群体。在一些实施方案中,抗原结合至固体支持物(例如,抗原共价或非共价结合至固相支持物)。在一些实施方案中,固体支持物可以是珠粒(例如,琼脂糖珠粒或磁珠)、柱的壁或板(例如,组织培养板)的底部。
在一些实施方案中,动物是以前暴露于抗原的动物。在一些实施方案中,以前暴露于抗原的动物是先前用抗原免疫的动物。
在另一个方面,本发明提供了用于获得特异性结合抗原的免疫球蛋白的免疫球蛋白链可变区的氨基酸序列的方法,其包括:(a) 提供编码动物的多种免疫球蛋白的免疫球蛋白可变区的核酸序列;(b) 获得特异性结合抗原的多克隆免疫球蛋白群体的免疫球蛋白链可变区的肽片段的质谱信息;(c) 将肽片段的质谱信息与核酸序列的预测质谱信息(所述预测质谱信息源自所述核酸序列编码的预测氨基酸序列)相关联,以获得包含所述肽片段的免疫球蛋白链可变区的氨基酸序列;和(d) 基于所述肽片段对所述可变区的氨基酸序列覆盖率从鉴定的免疫球蛋白链可变区的核苷酸序列或氨基酸序列中选择,以获得特异性结合抗原的免疫球蛋白可变区的核苷酸序列或氨基酸序列。
在一些实施方案中,所述方法进一步包括步骤(e)用免疫测定筛选所述免疫球蛋白链可变区的氨基酸序列,以分离特异性结合抗原的免疫球蛋白的免疫球蛋白链可变区。在一些实施方案中,在步骤(e)筛选步骤之前,通过重组分子生物学技术或基因合成技术合成步骤(d)中获得的免疫球蛋白链可变区的核苷酸序列或氨基酸序列。在一些实施方案中,将步骤(d)中产生的免疫球蛋白链可变区与第二免疫球蛋白链可变区装配以生成特异性结合抗原的免疫球蛋白的抗体结合结构域。在一些实施方案中,免疫测定选自流式细胞术测定、酶联免疫吸附测定(ELISA)、Western印迹测定、和免疫组织化学测定、免疫荧光测定、放射免疫测定、中和测定、结合测定、亲和力测定、或蛋白或肽免疫沉淀测定。
在一些实施方案中,免疫球蛋白链可变区是重链可变区或轻链可变区。
在进一步方面,本发明提供了用于生成特异性结合抗原的免疫球蛋白的抗原结合结构域的方法,其包括:(a) 提供编码来自动物的多种免疫球蛋白的免疫球蛋白重链可变区和轻链可变区的核酸序列;(b) 获得特异性结合抗原的多克隆免疫球蛋白群体的免疫球蛋白重链和免疫球蛋白轻链的肽片段的质谱信息;(c) 将肽片段的质谱信息与核酸序列的预测质谱信息(所述预测质谱信息源自所述核酸序列编码的预测氨基酸序列)相关联,以获得包含所述肽片段的免疫球蛋白链可变区的核苷酸序列或氨基酸序列; (d) 基于所述肽片段对所述可变区的氨基酸序列覆盖率从鉴定的免疫球蛋白链可变区的核苷酸序列或氨基酸序列中选择,以获得特异性结合抗原的免疫球蛋白的可变区的核苷酸序列或氨基酸序列;和(e) 将选择的免疫球蛋白重链可变区与选择的免疫球蛋白轻链可变区装配以生成特异性结合抗原的免疫球蛋白的抗原结合结构域。
在本发明的所有方面的各个实施方案中,动物是脊椎动物。在各个实施方案中,动物是哺乳动物。在一些实施方案中,动物是人。在一些实施方案中,动物是大鼠、兔或小鼠。在一些实施方案中,动物是鸟、驯养动物、伴侣动物、家畜动物、啮齿动物或灵长类动物。在一些实施方案中,动物是转基因非人动物,例如,表达人抗体序列和/或产生至少部分是人的抗体的转基因非人动物。
在各个方面,本发明还提供了根据本发明的各个非限制性实施方案分离或生成的特异性结合抗原的免疫球蛋白(或其可变区),或免疫球蛋白的免疫球蛋白链可变区或抗原结合结构域。在各个实施方案中,特异性结合抗原的免疫球蛋白(或其可变区),或免疫球蛋白的免疫球蛋白链可变区或抗原结合结构域是分离的或重组的。在各个实施方案中,本发明还提供了药学可接受的载体和根据本发明的各个非限制性实施方案分离或生成的特异性结合抗原的免疫球蛋白(或其可变区),或免疫球蛋白的免疫球蛋白链可变区或抗原结合结构域。
在进一步方面,本发明提供了治疗患有或怀疑患有特征在于疾病抗原的疾病的动物的方法,其中所述方法包括施用有效量的根据本发明的各个实施方案的组合物,其中组合物的免疫球蛋白(或其可变区)、或免疫球蛋白链可变区或抗原结合结构域特异性结合的抗原和疾病抗原是相同的。在一些实施方案中,动物是人。在一些实施方案中,动物是啮齿动物、家畜动物、驯养动物、伴侣动物或灵长类动物。
在进一步方面,本发明提供了降低动物中特征在于动物中存在疾病抗原的疾病的发生可能性的方法,其中所述方法包括施用有效量的根据本发明的各个实施方案的组合物,其中组合物的免疫球蛋白(或其可变区)、或免疫球蛋白链可变区或抗原结合结构域特异性结合的抗原和疾病抗原是相同的。在一些实施方案中,组合物进一步包含佐剂。在一些实施方案中,动物是人。在一些实施方案中,动物是啮齿动物、家畜动物、驯养动物、伴侣动物或灵长类动物。
附图简要说明
图1是包含两条重链和两条轻链的抗体的示意图。两条重链通过位于抗体铰链区的两个二硫键彼此连接。每条轻链经由单一二硫键连接至重链。在重链和轻链的N-末端处生成抗原结合位点。
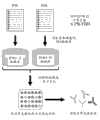
图2是显示本发明的各个实施方案的非限制性方法的一个实例的示意图。在该实例中,从相同动物(例如,人、小鼠或兔)收集包含B淋巴细胞的样品(例如,血液样品或组织样品)和血液血清和/或血浆。对编码免疫球蛋白链(或其可变区)的核酸分子进行测序,并且这些核酸序列用于基于由核酸序列编码预测的氨基酸序列而生成理论或预测的质谱信息。将来自血液血清的多克隆抗体蛋白水解消化或化学片段化,并且获得的肽片段通过质谱法进行分析。将来自核酸序列的信息(例如,质谱)与肽片段的质谱信息进行比较,以鉴定抗体的免疫球蛋白链(或其可变结构域)的序列。然后,可以根据标准方法重组生成该抗体。
图3是显示本发明的各个实施方案的非限制性方法的另一个实例的示意图。在该实例中,从相同动物(在这种情况下是兔)收集B淋巴细胞和血液血清和/或血浆。从B淋巴细胞,提取mRNA,并且使用免疫球蛋白基因特异性测序引物使用由454 Life Sciences商售的Genome Sequencer FLX System机器进行测序。该信息用于基于预测的氨基酸序列生成理论质谱。从血液血清和/或血浆,分离多克隆抗体,并且使用蛋白酶进行消化和/或使用化学蛋白切割试剂进行切割。获得的肽片段通过液相色谱法分离,随后进行质谱分析(MS/MS)。将肽片段的质谱与核酸序列的理论质谱相关联,以获得包括肽片段的免疫球蛋白链的氨基酸序列。然后可以通过将编码免疫球蛋白链的核酸序列克隆进表达载体中并且在细胞中表达该表达载体而将重链和轻链进行装配,以生成重组免疫球蛋白。然后进一步表征表达的重组免疫球蛋白。
图4是描述本发明的各个实施方案的非限制性方法的另一个实例的示意图。在该实例中,从相同动物(例如,人、小鼠或兔)收集非限制性的B细胞来源(例如,脾细胞)和多克隆抗体。核酸分子从B细胞来源提取,并使用Roche 454机器使用免疫球蛋白基因特异性测序引物进行下一代测序(NGS)。该信息(可以输入遗传物质数据库)可以用于基于由核酸序列编码的预测的氨基酸序列生成理论质谱。此外,将来自动物(例如,人、小鼠或兔)的多克隆抗体(或其肽片段)上样到质谱仪用于分析。使用Kabat规则分析核酸序列来鉴定序列的可变区(例如,CDR区或FR区之一)的序列。然后筛选来自分析的多克隆抗体的肽片段的序列,以鉴定哪些肽匹配来自预测的氨基酸序列的可变区的全部或部分。
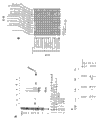
图5是显示在可变区具有良好质谱相关性和肽的重链和轻链NGS(即,下一代测序)序列的表。这些肽中的一些相当频繁出现(参见,例如,轻链,参考编号G623FKB01A3GC7)和一些具有高核酸序列频率计数(参见,例如,轻链,参考编号G623FKB01AXJ1C)。粗体斜体的行代表测试后被发现含有特异性结合抗原的序列的免疫球蛋白链(参见图6中的测试结果)。
图6是显示ELISA测定的结果的表,所述ELISA测定测试使用本发明的非限制性方法制备的抗体,所述抗体针对用p-Erk肽包被的ELISA板进行筛选。图5中显示的不同轻链和重链彼此随机结合。如从图6可见,一些配对导致产生能够特异性结合p-ERK-包被板的抗体(阳性肽以阴影显示)。
图7是琼脂糖凝胶的摄像图,其显示来自用p-MET抗原免疫的兔的脾细胞生成的cDNA的重链和κ和λ轻链的RT-PCR反应(即,逆转录酶-聚合酶链式反应)的结果。
图8是显示将源自核酸序列的理论(即预测)质谱与来自亲和纯化的抗体的LC-MS/MS数据组合后抗体链的序列。还显示基于NGS频率的抗体链丰度。合成斜体表示的链并装配成抗体;并且粗体斜体链是用Western印迹分析测试后被发现特异性结合p-MET抗原的链。
图9是显示Western印迹实验结果的示意图,其使用以本发明的非限制性方法生成的两种不同的兔抗体(印迹标记为1和2)和对照抗体(最左边的印迹)探测从未经处理的Hela细胞(所有三个印迹中的 - 道)或用人生长因子(HGF)处理的Hela细胞(所有三个印迹中的+道)制备的裂解物。用Western印迹检测到阳性结果后,然后鉴定抗原特异性抗体(重链和轻链配对)。如所示,泳道1和2两者中鉴定的抗体使用相同的重链,但具有不同的轻链。Western印迹结果下面显示两种兔抗体的重链和轻链的氨基酸序列,重和轻链的CDR3区用下划线表示。
图10a-e. 孕酮受体特异性多克隆兔IgG的亲和纯化。(a) 用蛋白A从免疫兔的血清中分离总IgG,并且进一步通过重力流在固定化的抗原肽上亲和纯化。充分洗涤以减少非特异性的IgG之后,渐进酸性pH的顺序洗脱用于将抗原特异性多克隆IgG分级。以匹配的抗体浓度(21.5ng/ml)通过Western印迹测试各级分的特异性活性以检测来自T47D细胞(+)的裂解物中的PR A/B。还测试来自HT1080 (-)的阴性对照裂解物。(b). 具有最高比活性的级分(pH1.8)用四种蛋白酶处理,用于LC-MS/MS分析。(c). 通过SEQUEST与含有 CDRH3(下划线)的V区全胰蛋白酶肽GFALWGPGTLVTVSSGQPK (SEQ ID NO:305)匹配的MS/MS谱,XCorr为5.560,且ΔM (观察的m/z – 预测的m/z)为0.39ppm。(d). MS/MS谱通过SEQUEST映射到V区肽,并且过滤至≤ 2%的FDR。然后将高置信度肽重新映射到通过NGS生成的V区数据库,考虑用于样品制备的蛋白酶并且保持记录肽的总数、独特肽的数量、谱共享和整个V区的氨基酸覆盖率。选择高覆盖率的V区序列,表达为单克隆抗体,并且筛选所需活性。(e). 克隆F9的重链和轻链序列鉴定覆盖率。描述的V区序列,当配对时,特异性结合人PR A/B(参见图11a-e)。由一种或多种肽映射的氨基酸以粗体显示。为了使V区覆盖率最大化和负责高度可变的氨基酸组成,使用互补的蛋白酶(糜蛋白酶、弹性蛋白酶、胃蛋白酶、胰蛋白酶。
图11a-e. 针对孕酮受体A/B的功能性单克隆抗体的鉴定和表征。(a). 重链和轻链的组合配对产生了以黄色表示的12个抗原特异性ELISA反应克隆。CDR3序列用作标识符: 表示Western印迹阳性克隆(参见图11b)。(b). 6个克隆(F1 F9、H1、C1、F7、和H9)对于通过Western印迹的孕酮受体A/B检测是特异性的。克隆E6(ELISA阴性,Western阴性)和H7(ELISA阳性,Western阴性)显示为对照。+,T47D (PR A/B-阳性);-,MDA-MB-231 (PR A/B-阴性)。所有抗体在21.5 ng/mL进行测试。(c). 通过免疫组织化学比较克隆F9与亲和纯化的多克隆混合物的比活性。0.4 ug/mL F9特异性染色PR A/B-阳性组织或细胞系(T47D和MCF-7),但没有特异性染色PR A/B-阴性细胞系(MDA-MB-231)。0.2 μg/mL多克隆抗体用作阳性对照。(d). 流式细胞术分析。蓝色,T47D细胞(孕酮受体A/B阳性细胞系);黑色,MDA-MB-231(孕酮受体A/B阴性细胞系)。多克隆抗体信/噪比,1.69;浓度,3.7μg/mL。单克隆抗体F9信/噪比= 36.4;浓度0.5μg/mL。(e). 共聚焦免疫荧光显微镜分析显示在0.46μg/ mL时对于孕酮受体A/B阳性细胞系MCF-7,但不对MDA-MB-231细胞的特异性核染色模式。没有包括一抗作为背景染色对照。多克隆抗体还用作在1.85μg/mL浓度时的比较。
图12a-d. 克隆C3抗Lin28A单克隆抗体的表征。(a) 重链和轻链的组合配对产生了以阴影表示的5个抗原特异性ELISA反应克隆。√表示Western印迹阳性克隆。CDR3序列用作标识符。(b) 使用各种Lin28A阳性细胞裂解物NCCIT、NTERA、mMES和IGROV1进行Western印迹分析。(c) 使用Lin28A阴性细胞(HeLa)和Lin28A阳性细胞(NTERA)进行共聚焦免疫荧光分析。(d) 单克隆抗体的流式细胞术分析。左峰,HeLa细胞(Lin28A - );右峰,NTERA细胞(Lin28A +)。* V区具有相同的CDR3序列但不具有相同的V区序列。
图13a-c. 针对磷酸化-Erk的功能性小鼠单克隆抗体的鉴定和表征。(a) 从三只小鼠的合并血清纯化磷酸化-Erk多克隆抗体。通过Western印迹测定合并血清、来自合并血清的蛋白G纯化的总IgG、来自蛋白G纯化的未结合的级分、和pH3.5、2.7和1.8的酸洗脱级分针对Jurkat细胞裂解物中的磷酸化-Erk的结合特异性。+,用TPA刺激的Jurkat细胞; - ,用U0126处理的Jurkat细胞。(b) 重链和轻链的组合配对产生了以阴影表示的15个通过肽抗原ELISA有反应性的克隆。√表示Western印迹阳性克隆(参见(c))。CDR3序列用作标识符。对于重链序列,下划线部分表示构架区3的最后。(c) 三个克隆(C10、F10和M3)通过Western印迹对磷酸化-Erk检测是特异性的。克隆C9(ELISA阳性,Western阴性)显示为对照。所有抗体在100 ng/mL进行测试。
详细描述
本公开涉及用于迅速且准确获得特异性结合目标抗原的单克隆抗体的氨基酸序列(和编码核酸序列)的方法和系统。更具体地,本发明方法涉及针对遗传物质数据库直接的、基于质谱的蛋白组学研究来自动物血清的循环多克隆抗体,所述遗传物质数据库由编码全长免疫球蛋白链或可变区的核酸分子构成。在具体实施方案中,遗传物质数据库通过利用核酸测序技术从动物(例如,收集其血清以获得多克隆抗体的相同动物)的B细胞所有组成成分生成。因此,本方法基本上涉及关联(即交叉比较或交叉引用)来自两个来源的信息:来自动物的实际循环多克隆抗体的质谱信息,和来自遗传物质数据库的信息(包括,例如,预测质谱)。然后可以从对应于实际血清抗体的遗传物质数据库鉴定重链和轻链DNA序列的列表。可以成对表达这些重链和轻链以获得功能性单克隆抗体。
在一些实施方案中,本发明方法不需要B细胞永生化、单细胞分选和分子克隆、或噬菌体显示,并且不涉及基于猜测装配抗体序列。通过利用质谱技术和核酸测序技术(诸如下一代DNA测序或NGS)的力量,本发明方法可以显著地减少从多克隆群体分离抗原特异性的单克隆抗体序列所需的时间量,从而能更迅速地转换至可以治疗使用的重组抗体诸如完全人抗体或人源化抗体(例如人源化鼠抗体)。
此外,本发明方法能够鉴定现有技术可能错过的稀有抗体。本发明人令人惊讶地发现,具有非常选择性特异性的个别抗体(例如,特异性结合多肽内的磷酸化酪氨酸残基的抗体)可以非常稀少地存在于多克隆群体内。依靠编码抗体的mRNA的频率和PCR扩增的方法可能错过这些抗体,因为它们的可变链以低频存在。相比之下,本发明方法利用,例如,源自多克隆抗体群体的实际肽片段的基于质谱的蛋白组学分析,因此不会受到PCR扩增后频率误差的影响。
此外,本方法允许快速生成可能在开始的多克隆群体中不存在的新的抗原特异性抗体。例如,生成的具有最高期望质量(例如,对于抗原或所需同种型(例如,IgG2a)具有最高结合亲和力(或最低KD))的免疫球蛋白分子可能是由于来自多克隆群体中第一抗体的轻链与多克隆群体中第二抗体(即,不同于第一抗体)的重链装配在一起的结果。
本文描述的方法可用于基础免疫学和治疗中。例如,所述方法可以为理解免疫学领域的核心问题(包括抗原暴露后血清抗体多样性、动力学(dynamics)、动力学(kinetics)、克隆性和B细胞的迁移)提供基础。所述方法也可以用于从免疫、自然感染或患病个体寻求治疗相关的人单克隆抗体。
如本文表明,本方法已成功地应用于实验室动物物种和人两者中的几种不同抗原,并且导致分离具有抗原特异性活性的单克隆抗体,所述抗原特异性活性囊括或超过在经免疫受试者血清中发现的初始亲和纯化的多克隆抗体的抗原特异性活性。
因此,本公开进一步提供了对于抗原特异性的分离的重组单克隆抗体,包括对于疾病抗原特异性的治疗性抗体,以及用于基于施用治疗性单克隆抗体治疗疾病的治疗方法。
下面更详细地描述了本发明的各个方面和实施方案。本文中提到的专利、公开的申请和科学术语确立了本领域技术人员的知识,且完整地通过引用并入本文,其程度等同于各自被特定且单独地通过引用并入。本文引用的任何参考文献和本说明书中的具体教导之间的任何冲突应当以有利于后者的方式来解决。同样,词或短语的本领域理解的定义和本说明书中具体教导的词或短语的定义之间的任何冲突应当以有利于后者的方式来解决。
定义
如本文所使用的,以下术语具有指定的含义。说明书中所用的单数形式“一(a)”、“一(an)”和“该(the)”特别也包括它们所引用的术语的复数形式,除非内容另有明确指出。术语“约”用于本文中是指接近,在该区间内,大致,或左右。当术语“约”连同数值范围使用时,其通过延伸所述数值上面和下面的边界而修饰该范围。通常,术语“约”在本文中用于以20%的方差修饰所述值上方和下方的数值。
“肽”或“肽片段”是指从将个体氨基酸残基连接在一起而形成的短聚合物,其中一个氨基酸残基和第二个氨基酸残基之间的连接被称为酰胺键或肽键。肽包含至少两个氨基酸残基。肽与多肽的区别在于肽较短。通过一个肽的C'末端氨基酸残基和第二个肽的N'末端氨基酸残基之间的酰胺键或肽键连接在一起的至少两个肽形成根据本发明的各个实施方案的多肽。
“多肽”是指从连接个体氨基酸残基形成的长聚合物,其中一个氨基酸残基和第二个氨基酸残基之间的连接被称为酰胺键或肽键。多肽包含至少四个氨基酸残基;然而,多个多肽可经由酰胺或肽键连接在一起以形成一个甚至更长的多肽。
“核酸分子”是指从将个体核苷酸(例如,脱氧核糖核苷酸或核糖核苷酸)连接在一起而形成的聚合物,其中一个核苷酸和另一个核苷酸之间的连接是共价键,包括,例如,磷酸二酯键。因此,术语包括但不限于DNA、RNA和DNA-RNA杂合体。
“核酸序列”是指包括编码全部或部分免疫球蛋白链(例如,重链或轻链)的核苷酸的核酸序列(或与其互补的核苷酸序列)。在一些实施方案中,核酸序列是基因组DNA(例如,具有或不具有内含子DNA的外显子DNA)。在一些实施方案中,核酸序列是基因组cDNA或RNA的一些形式(例如,hnRNA、mRNA等)。在一些实施方案中,核酸序列是表达的核酸序列,所述序列在含有该核酸序列的细胞中将转录成核酸分子(例如,DNA转录成RNA)或翻译成多肽。同时,表达核酸分子包括但不限于hnRNA、mRNA、cDNA和基因组外显子序列。在核酸分子方面的“互补”简单是指两个单链核酸分子含有将形成标准Watson-Crick碱基对以形成双链核酸分子的核苷酸,无论双链分子是DNA、RNA或DNA-RNA杂合体。
如本文所使用的,“B淋巴细胞”是指其中基因重组(或基因重排)已经开始在含有编码免疫球蛋白链的基因的基因座发生的任何白血细胞。例如,人免疫球蛋白基因存在于染色体14(重链基因座)、染色体2(κ轻链基因座)和染色体22(λ轻链基因座)。如果人白血细胞在免疫球蛋白链基因座(例如,染色体14,染色体2,或染色体22)中已经经历了基因重排事件,则该细胞被认为是B淋巴细胞。因此,B淋巴细胞包括但不限于,B细胞、pre-B细胞、pro-B细胞包括早期pro-B细胞(例如,其中重链基因的D和J区已经经历重排,但轻链基因是种系的(即,没有重排))和晚期pro-B细胞(例如,其中重链基因的V、D和J区重排,但轻链基因仍然是种系的并且其中没有免疫球蛋白蛋白表达于在细胞表面上)、pre-B细胞包括大pre-B细胞和小pre-B细胞、未成熟的B细胞、活性B细胞、生发中心B细胞、浆细胞(包括浆母细胞)和记忆性B细胞。
在整个说明书和权利要求中,术语“抗体”和“免疫球蛋白”可以互换使用,意指包括任何同种型或亚同种型的完整免疫球蛋白多肽分子(例如IgG、IgG1、IgG2、IgG2a、IgG2b、IgG3、IgG4、IgM、IgD、IgE、IgE1、IgE2、IgA),其来自以下任何动物物种,诸如灵长类动物(例如人或黑猩猩)、啮齿类动物(例如小鼠或大鼠)、兔形目动物(例如家兔或野兔)、牲畜动物(例如牛、马、山羊、猪和绵羊)、鱼(例如鲨鱼)、鸟类(例如鸡)或骆驼科动物(例如,骆驼或美洲驼),或来自基因工程改造以产生人抗体的转基因非人动物(例如啮齿类动物)(参见,例如,Lonberg等人, WO93/12227; 美国专利号5,545,806; Kucherlapati,等人, WO91/10741; 美国专利号. 6,150,584; US 2009/0098134; US 2010/0212035; US 2011/0236378; US 2011/0314563; WO2011/123708; WO2011/004192; WO2011/158009);其抗原结合结构域片段,诸如Fab、Fab'、F(ab')2;其变体诸如scFv、Fv、Fd、dAb、双特异性scFvs、双抗体、直链抗体(参见美国专利号5,641,870;Zapata等人, Protein Eng. 8 (10):1057-1062.1995);单链抗体分子;和由抗体片段形成的多特异性抗体;和包含是抗体结合结构域(本文中其他地方定义)或与其同源的结合结构域的任何多肽。本发明的各个实施方案的非限制性抗体包括,但不限于多克隆抗体、单克隆抗体、单特异性抗体、多特异性抗体和其片段、包含融合至另一个多肽的免疫球蛋白结合结构域的嵌合抗体和人源化抗体诸如非人抗体(例如,兔抗体),其恒定和/或FR结构域已经被来自人抗体的恒定和/或FR结构域所替代(参见,例如,美国专利号5,530,101; 5,585,089; 5,693,761; 5,693,762; 6,180,370; 和6,548,640)。基因工程改造以产生人(例如,至少是部分人)抗体的转基因非人动物可以从以下获得:Harbour Antibodies (Rotterdam, The Netherlands)、Ablexis (San Francisco, CA), Kymab Ltd (Cambridge, UK)、OMT, Inc. (Palo Alto, CA), Amgen (Thousand Oaks, CA), Medarex (Princeton, NJ),和Regeneron (Tarrytown, NY)。
天然存在的完整的抗体是由两类多肽链轻链和重链组成。本发明各个方面的非限制性抗体可以是包含两条重链和两条轻链的完整的四条免疫球蛋白链抗体。抗体的重链可以是任何同种型(包括IgM、IgG、IgE、IgA或IgD)或亚同种型(包括IgG1、IgG2、IgG2a、IgG2b、IgG3、IgG4、IgE1、IgE2等)。轻链可以是κ轻链或λ轻链。例如,单一IgG天然存在的(或完整的)抗体包含两个相同拷贝的轻链和两个相同拷贝的IgG重链。所有天然存在的抗体的重链(其中每条重链含有一个可变结构域(VH)和一个恒定结构域(CH,其本身包含CH1区、铰链区、CH2区和CH3区))彼此经由其恒定结构域内的多个二硫键彼此结合以形成抗体的“茎”。所有天然存在的抗体的轻链(其中每条轻链含有一个可变结构域(VL)和一个恒定结构域(CL))各自通过其恒定结构域经由二硫键结合而结合至一个重链恒定结构域。四条免疫球蛋白链抗体(例如IgG抗体)的示意图显示在图1中。在图1中,三个CH结构域以淡蓝色显示,单一VH结构域以深蓝色显示,单一CL结构域以淡粉色显示,并且单一VL结构域以深粉色显示。如图1中所示,轻链和重链的VL和VH结构域分别来到一起形成抗体结合结构域。
在一些实施方案中,完整免疫球蛋白链(例如,重链或轻链)可以5'至3'的顺序(对于编码该链的核酸序列)或氨基末端至羧基末端的顺序(对于该链的氨基酸序列)包含:可变结构域和恒定结构域。可变结构域可以包含3个互补决定区(CDR;也称为高变区或HVs),具有间插的构架(FR)区。轻链和重链两者的可变结构域含有在四个更保守构架区(FR)之间夹着的3个高变区,结构为5' (或N')- FR1、CDR1、FR2、CDR2、FR3、CDR3、FR4 3' (或C'),从恒定区3'(或C')至FR4区。CDR形成环,所述环包含抗体的主要抗原结合表面(参见Kabat, E. A.等人, Sequences of Proteins of Immunological Interest, National Institutes of Health, Bethesda, Md., (1987)和Wu, T.T.和Kabat, E.A. (1970) J. Exp. Med. 132:211-250 (1970)),其中四个构架区主要采用β-折叠构象和CDRs 形成环,所述环连接、在某些情况下形成β折叠结构的一部分。每条链上的CDR和其他链的CDR被近处的构架区束缚,形成抗原结合结构域。
“抗原”是指可以被抗体特异性结合的目标分子(例如,多肽或碳水化合物)。被抗体特异性结合的抗原部分被称为“表位”。“表位”是能够被抗体的抗原结合结构域特异性结合的目标分子的最小部分。表位的最小大小可以是约三至七个氨基酸(例如,5个或6个氨基酸)。单一抗原上可能有多个表位,因此,单一抗原可以被多种不同抗体特异性结合,所有这些抗体都特异性结合抗原(即,所有这些抗体是抗原特异性抗体),甚至尽管每个个体抗体特异地结合到抗原上不同表位。
“疾病抗原”是指在疾病状态过程中在动物中产生的抗原。例如,病毒抗原(例如,由病毒的遗传物质的核酸分子编码的抗原)是被该病毒感染的动物中的疾病抗原。同样,一些疾病(例如癌症)的特征在于产生嵌合蛋白(例如,BCR-ABL)的基因易位。因此,BCR-ABL蛋白是疾病抗原。应当理解的是,疾病抗原不一定仅见于患有该种疾病的动物中。
“疾病”简单地是指影响动物的任何异常状况。疾病的非限制性实例包括但不限于:自身免疫性疾病(例如,风湿性关节炎或I型糖尿病),癌症(例如,白血病、结肠癌或前列腺癌等),病毒感染(例如,由HIV病毒感染引起的AIDS或由水痘带状疱疹病毒感染引起的鸡水痘),寄生虫感染(例如,血吸虫病或疥疮)和细菌感染(例如,肺结核或白喉)。
“特异性结合”是指免疫球蛋白或抗体与其抗原(即,其特异性抗原)相互作用,其中相互作用依赖于抗原上的特定结构(例如,表位)的存在;换言之,抗体识别并结合至特定结构,而不是所有一般分子或结构。特异性结合抗原的抗体可以被称为“抗原特异性抗体”或“对于抗原特异性的抗体”。在一些实施方案中,特异性结合抗原的抗体可以从含有该抗原以及其他分子的溶液(例如,细胞裂解物)中免疫沉淀该抗原。在一些实施方案中,特异性结合其抗原的抗体对于其抗原具有1x 10-6 M或更小的KD。在一些实施方案中,特异性结合其抗原的抗体对于其抗原具有1 x10-7 M或更小的KD、或1 x10-8 M或更小的KD、1 x 10-9 M或更小的KD、1 x 10-10 M或更小的KD、1 x 10-11 M或更小的KD、1 x 10-12 M或更小的KD。在某些实施方案中,特异性结合其抗原的抗体对其特异性抗原的KD为1 pM至500 pM、或500 pM至1 μM、或1 μM至100 nM、或100 mM至10 nM。如本文所使用的,术语“KD”意指两个分子之间的相互作用的解离常数(例如,抗体和其特异性抗原之间的解离常数)。
“免疫球蛋白链的可变区”或“免疫球蛋白链可变区”是包含免疫球蛋白的重链(即,VH结构域)或轻链(即,VL结构域)的可变结构域的至少一部分的多肽,其中VL和VH结构域的部分形成免疫球蛋白的抗原结合结构域(参见图1)。因此,免疫球蛋白的可变区可以包括,但不限于,单一CDR(例如,CDR1),间插单一FR的两个CDRs(例如,CDR1、FR2、和CDR2)、间插两个FR的3个CDRs(例如,CDR1、FR2、CDR2、FR3、和CDR3)、或侧翼为FR1和FR4中任一种或两者的3个CDRs(例如,FR1、CDR1、FR2、CDR2、FR3、CDR3、FR4)。在一些实施方案中,免疫球蛋白链可变区是重链或轻链之一上的区域,当与完整免疫球蛋白的另一条链(即,轻链或重链)的免疫球蛋白链可变区相组合时,其形成抗原结合结构域。
“抗原结合结构域”是指免疫球蛋白中单一重链与单一轻链装配的区域,其保留完整抗体对其特异性抗原的特异结合活性。因此,包含两条重链和两条轻链的完整IgG免疫球蛋白具有两个抗原结合结构域。同样,保留重链和轻链之间的共价键的完整抗体的片段化也将导致产生具有抗原结合结构域的免疫球蛋白片段。例如,用酶木瓜蛋白酶消化免疫球蛋白将生成F(ab)片段,其中每一个具有单一抗原结合结构域。当然,整个F(ab)不是抗原结合结构域;反而只有保留特异性结合抗原的能力的F(ab)片段部分是抗原结合结构域。
获得抗原特异性免疫球蛋白序列的方法
在一个方面,本发明涉及用于从多克隆抗体群体获得单一免疫球蛋白的免疫球蛋白链(或其可变区)的氨基酸和/或核酸序列的方法。
根据本发明的方法,目标多克隆抗体群体从动物获得,且片段化以生成肽片段,所述肽片段通过质谱进行分析。然后,将从肽片段观察到的质谱信息与源自遗传物质数据库的预测质谱信息相关联,所述遗传物质数据库由编码全长免疫球蛋白重链和/或轻链(或其可变区)的核酸序列构成。作为这样的关联的结果,免疫球蛋白重链和/或轻链(或其可变区)可以从遗传物质数据库鉴定,所述遗传物质数据库对应于开始多克隆抗体群体内的免疫球蛋白分子的免疫球蛋白重链和/或轻链(或其可变区)。
下面更详细地描述了本方法的各个方面。
多克隆抗体的开始群体
特异性结合目标抗原的免疫球蛋白可以从动物(其包括任何哺乳动物,诸如人)收集。免疫球蛋白可以从动物的体液样品收集,所述体液样品包括,例如,血液、血清或血液的血浆、脑脊液、滑液、腹膜液、粘膜分泌物、泪液、鼻分泌物、唾液、乳和泌尿生殖道分泌物。
在一些实施方案中,免疫球蛋白不需要来自单一个体动物,但,反而可能是取自不同个体的不同抗体(单克隆抗体或多克隆抗体)的混合物。在一些实施方案中,免疫球蛋白从转基因非人动物,例如,表达人抗体序列和/或产生至少部分是人的抗体的转基因非人动物收集。
在一些实施方案中,这些免疫球蛋白对于目标抗原是特异性的,因为由其收集免疫球蛋白的动物以前用抗原免疫,或因为由其收集免疫球蛋白的动物以前暴露于动物可能生成抗原特异性抗体的状况。在后者情况的一个实例中,动物可能已被病毒(如EB病毒)感染,其中目标抗原是由EB病毒的基因组编码的EBNA1蛋白。
在各个实施方案中,收集(即,获得)其免疫球蛋白的动物是与收集其B淋巴细胞核酸序列以生成参考数据库的动物是相同物种。在一些实施方案中,收集其免疫球蛋白用于肽数据库的动物与收集其B淋巴细胞核酸用于参考数据库的动物是相同的动物。
如图2中显示,当所述动物是相同的动物时,取自动物的血液可以提供核酸序列(例如,来自血液中的细胞)和多克隆抗体(例如,来自血液的血清或血浆)两者。
从动物收集的免疫球蛋白形成免疫球蛋白的多克隆群体,因为不同的B淋巴细胞产生群体的成员。应当指出,在这样的多克隆群体中,不是该多克隆群体内的所有个别抗体都将特异性结合相同抗原。事实上,群体内的每种抗体可以结合不同的抗原。然而,该多克隆群体仍然被称为特异性结合具体抗原,如果多克隆群体的至少一种个别抗体,优选多种抗体,结合抗原(参见,例如,下面的实施例3)的话。在另一个实例中,多克隆群体内的一些抗体可以以低亲和力结合抗原。然而,多克隆群体被称为特异性结合抗原,如果该群体中的一些(例如,至少一种或多种)特异性结合抗原的话。
应当指出,短语“特异性结合抗原的多克隆抗体(免疫球蛋白)”是指,多克隆群体内,至少一种抗体特异性结合抗原,然而,所述一种抗体不必与多克隆群体内不特异性结合抗原的其他抗体相分离。当然,在一些实施方案中,多克隆群体内多于一种不同的抗体特异性结合抗原。
还应当指出,不同的抗体分子是由不同的B细胞产生的抗体分子。例如,在收集血清之后,1000个抗体分子的多克隆群体可以从血清中分离出 (例如,使用抗体结合至蛋白A柱以便将抗体与其他血清成分相分离)。在该1000个抗体分子的群体内,900个可能是相同的(即,由相同的B细胞分泌),因此,该多克隆群体内真正只存在101种不同的抗体。关于多克隆群体,如果所有900个相同的抗体分子特异性结合抗原,则1000个抗体分子的多克隆群体是特异性结合抗原的多克隆抗体。类似地,如果剩余的100个不同抗体分子中额外5个不同的抗体分子特异性结合抗原,则1000个抗体分子的多克隆群体同样是特异性结合抗原的多克隆抗体。
多克隆群体内的大多数抗体分子不必特异性结合抗原,因为该群体被称为“特异性结合抗原的多克隆抗体”。例如,如果在1000个抗体分子的多克隆群体内,即使只有1个抗体分子特异性结合抗原且999个抗体分子不特异性结合抗原,则1000个抗体分子的群体仍然是如本文中使用的术语“特异性结合抗原的多克隆抗体”。
还注意,多克隆抗体群体中的所有抗体不必结合抗原上相同的表位。例如,当群体内的每种不同抗体特异性结合抗原上不同的表位时,多克隆群体对于抗原可以是特异性的。
在本发明的非限制性方法的各个实施方案中,多克隆免疫球蛋白的群体可能具有,例如,群体内的至少两种不同的免疫球蛋白,或群体内的至少三种,或至少五种或至少10种,或至少20种或至少50种,或至少100种或至少500种不同的免疫球蛋白。
本发明还考虑从体外生长的B细胞的组织培养上清液收集免疫球蛋白的多克隆群体(例如,其中核酸序列从B细胞本身收集)。例如,B细胞群体可以从已经受EB病毒感染的动物收集。群体可以扩增,例如,以便与其他白血细胞相比富集群体中的B淋巴细胞。从这些细胞的该经培养的培养基(多克隆抗体由细胞分泌到其中)可以分离抗体的多克隆群体。
首先可以纯化从一种或多种动物或B细胞的组织培养上清液收集的免疫球蛋白的多克隆群体,然后消化成肽片段。例如,收集的多克隆抗体可以加入蛋白A或蛋白G琼脂糖柱,所述蛋白A或蛋白G琼脂糖柱可以将抗体与(例如)其他血清蛋白相分离。参见,例如,图2和图3。可替代地或额外地,收集的多克隆抗体进行抗原亲和纯化,以富集具有高比活性的抗体。尽管不是完全必要的,但是纯化步骤,尤其是抗原亲和纯化,可以减少多克隆混合物的复杂性,且最终降低潜在假阳性或假阴性候选免疫球蛋白的数量。收集的多克隆抗体可以在纯化之前和/或纯化之后进行浓缩和/或缓冲液交换。
在一个说明性实施方案中,为了从动物收集特异性结合目标抗原的免疫球蛋白,从动物采集外周血,且根据标准方法(例如,将抗体结合至蛋白A)收集血清和/或血浆抗体。血清和/或血浆抗体然后进行纯化或筛选,以富集特异性结合抗原的免疫球蛋白。该筛选可以是,例如,通过用抗原包被固相表面(例如,琼脂糖珠粒或塑料板的底部)并且在特异性结合抗原的免疫球蛋白将结合的条件下使血清和/或血浆通过抗原包被的表面。结合的抗体可以用蛋白酶(例如木瓜蛋白酶)或化学蛋白切割试剂(其特异性切割免疫球蛋白铰链区附近,且去除非结合的Fc部分)处理。洗掉未结合的血清和/或血浆蛋白(包括非特异性免疫球蛋白)之后,可以收集抗原特异性免疫球蛋白,并且因此与不特异性结合抗原的抗体相比富集它们的量。
从收集的多克隆抗体观察到的质谱
为了获得观察的(即,实际的)质谱,通过蛋白分析方法(例如,质谱法、液相色谱法等)分析收集的多克隆抗体(或其片段)。
在一些实施方案中,观察到的质谱信息从由多克隆抗体生成的肽片段获得。多克隆抗体可以进行片段化,例如,用一种或多种蛋白酶,和/或化学蛋白切割试剂,诸如溴化氰。
已知某些蛋白酶在特异性位点切割它们的底物。表1提供了常用的蛋白酶和它们的切割位点(以3字母氨基酸代码表示)的非全面的列表。
表 1
| 蛋白酶 | 切割位点 |
| 胰蛋白酶 | Arg或Lys(即在其羧基侧)之后切割,除非随后为Pro |
| 糜蛋白酶 | Phe、Trp或Tyr之后切割,除非随后为Pro |
| 弹性蛋白酶 | Ala、Gly、Ser或Val之后切割,除非随后为Pro. |
| 胞内蛋白酶Lys-C | Lys之后切割 |
| 胃蛋白酶 | Phe或Leu之后切割 |
| 嗜热菌蛋白酶 | Ile、Met、Phe、Trp、Tyr或Val之前切割,除非随后为Pro. |
| 胞内蛋白酶V8 (别名Glu-C) | Glu之后切割 |
可以用于将蛋白消化成更小片段的蛋白酶的更全面列表在Riviere和Tempst 的表11.1.1和11.1.3中给出(Riviere LR, Tempst P. Enzymatic digestion of proteins in solution. Curr Protoc Protein Sci. 2001 May; Chapter 11:Unit 11.1. PubMed PMID: 18429101; 整体通过引用并入本文)。
在具体实施方案中,多种(即,两种或更多种)蛋白酶用于(例如,单独或一起)消化多克隆抗体,以便使V区覆盖率最大化且导致免疫球蛋白的高度可变的氨基酸组成。例如,可以使用糜蛋白酶、弹性蛋白酶、胃蛋白酶和胰蛋白酶的组合,如本文中实施例7中显示。在一些实施方案中,基于遗传物质数据库中的核酸分子的分析根据一种或多种蛋白酶在预测的CDR3区内不切割而对其进行选择。
蛋白可以通过用特定化学蛋白切割试剂而非蛋白水解酶处理而被消化成可以易于通过质谱分析的更小的片段。参见例如G. Allen. Sequencing of Proteins and Peptides, Laboratory Techniques in Biochemistry and Molecular Biology, Vol. 9. Elsevier 1989的第三章。这些化学蛋白切割试剂包括但不限于,溴化氰、BNPS-粪臭素、亚碘酰苯甲酸、稀酸(例如稀盐酸),等等。例如,蛋白可以用溴化氰在Met残基处切割,氰基化后在Cys残基处切割,用BNPS-粪臭素或亚碘酰苯甲酸在Trp残基处切割等。蛋白片段也可以通过暴露于稀酸(例如,HCl)而生成。Zhong等人给出了使用部分酸水解来通过质谱法确定蛋白序列的一个实例 (Zhong H,等人, J. Am. Soc. Mass Spectrom. 16(4):471-81, 2005. PubMed PMID: 15792716, 整体通过引用并入)。Zhong等人同上使用用水中的25%三氟乙酸的微波辅助酸水解来片段化细菌视紫红质,用于通过质谱法进行测序。还参见Wang N,和Li L., J. Am. Soc. Mass. Spectrom. 21(9):1573-87, 2010.PubMed PMID: 20547072 (整体通过引用并入本文)。
蛋白可以进行片段化,以使它们更适合用于通过一种蛋白酶处理,通过组合的多于一种蛋白酶处理,通过用化学切割试剂处理,或通过组合的多于一种化学切割试剂处理,或通过组合的蛋白酶和化学切割试剂处理的质谱。该反应可以在升高的温度或升高的压力下进行。参见例如López-Ferrer D, 等人, J. Proteome. Res. 7(8):3276-81, 2008. PubMed PMID: 18605748; PubMed Central PMCID: PMC2744211 (整体通过引用并入)。可以允许片段化去完成,使得蛋白在所有键处被切割,以至于消化试剂能够切割;或者可以对消化条件进行调整,使得片段刻意不去完成,以产生较大片段,所述较大片段特别有助于解码抗体可变区序列;或可以对消化条件进行调整,使得蛋白部分消化成结构域,例如,如同用大肠杆菌DNA聚合酶I进行以制备Klenow片段。可以改变以调节消化水平的条件包括持续时间、温度、压力、pH、不存在或存在蛋白变性剂、特定蛋白变性剂(例如尿素、盐酸胍、去污剂、酸切割性去污剂、甲醇、乙腈、其他有机溶剂)、变性剂浓度,切割试剂的量或浓度或其与待消化蛋白的相对重量比等等。
在一些实施方案中,用于切割蛋白的试剂(即,蛋白酶或化学蛋白切割试剂)是完全非特异性试剂。使用这种试剂,对于在肽的N-末端、肽的C-末端或N-末端和C-末端两者起作用没有限制。例如,被限制在肽序列的一端或另一端、但非两端同时具有胰蛋白酶切割位点的部分蛋白水解的序列可用于本文描述的各种方法中。
获得的肽片段可以使用与由其生成观察到的质谱的质谱仪偶联的HPLC进行检测和分析。该方法可以被称为“自下向上(bottom up)”蛋白组学方法,其中将蛋白还原成相对小的肽(例如,3-45个残基长度)之后分离和鉴定蛋白质组组分。
在其他实施方案中,一种替代的 “自上向下(top down)”蛋白组学方法可以用于获得观测到的质谱,其中包括完整蛋白或大蛋白片段或蛋白结构域或大多肽的质谱分析。例如,为了鉴定将特异性抗原识别赋予特定多克隆抗体分子的抗体可变区的部分,通过直接分析足够大以使CDR保持连接在一起的片段而对可变区的大部分测序以确定其CDR是有帮助的。
对于描述“自下向上”和“自上向下”策略的综述,参见Han X, Aslanian A, Yates JR 3rd. Mass spectrometry for proteomics. Curr Opin Chem Biol. 2008 Oct;12(5):483-90. 综述. PubMed PMID: 18718552; PubMed Central PMCID: PMC2642903 (整体通过引用并入)。对于应用于确定抗体序列的自上向下蛋白质组学的新近综述,参见Zhang Z.等人, Mass Spectrom Rev. 2009 Jan-Feb;28(1):147-76. 综述. PubMed PMID: 18720354 (整体通过引用并入)。对于显示通过自上向下蛋白质组学对单克隆抗体的广泛测序的新近综述,参见Tsybin等人, Anal Chem. 2011 Oct 21. PubMed PMID: 22017162 (整体通过引用并入)。
在上述非限制性方法的一些实施方案中,当抗原特异性免疫球蛋白结合到抗原包被的表面时,免疫球蛋白可以分别用木瓜蛋白酶或胃蛋白酶消化以生成F(ab)和F(ab)2片段。由于免疫球蛋白链可变区的全部位于F(ab)片段的链上,这种用木瓜蛋白酶和/或胃蛋白酶预处理将富集免疫球蛋白链可变区。洗掉免疫球蛋白的非结合部分之后,可以收集免疫球蛋白链可变区。
免疫球蛋白片段通过质谱仪之后,将生成许多观察到的质谱。然而,鉴于多克隆群体内可能存在大量不同的免疫球蛋白(各自具有不同的氨基酸序列,其用质谱仪分析),获得的观察到的质谱将难以装配回功能性免疫球蛋白链可变区。在本发明的各个实施方案的方法中,因为基础核酸序列是可用的,所以不必装配观察到的质谱数据。相反,可以将观察到的单一肽片段的质谱与核酸序列的预测的质谱相关联,以便从开始多克隆免疫球蛋白群体获得特异性结合抗原的免疫球蛋白的整个免疫球蛋白链(或其可变区)的氨基酸(和基础核苷酸)序列。本文下面进一步说明该关联步骤。
除质谱信息以外,源自多克隆抗体的肽片段的额外信息可用于本发明的各个实施方案中。该信息包括但不限于,每种肽的质量,每种肽的长度(以氨基酸残基为单位),观察到的每种肽的质谱图(例如,来自串联质谱法诸如MS2或MS3谱),每种肽的质荷比,每种肽的离子电荷,每种肽的色谱概况,和每种肽的氨基酸序列。
质谱分析
在本发明的方法中,质谱信息可以通过质谱分析收集的免疫球蛋白或由其生成的片段而获得。质谱仪是一种能够测量个别电离化分子的质荷比(m/z)的仪器,其使研究人员能够鉴定未知化合物以定量已知化合物,并阐明分子的结构和化学特性。在一些实施方案中,开始通过将样品分离和上样到仪器上而开始质谱分析。一旦上样,样品被蒸发,然后电离。随后,离子将经由暴露于磁场根据其质荷比进行分离。在一些实施方案中,使用一个扇区仪器(sector instrument),并且根据当离子通过仪器的电磁场时离子轨迹的偏转幅度(这与离子质荷比直接相关)定量离子。在其他实施方案中,当离子通过四极管时,或基于它们在三维或线性离子阱或轨道阱(Orbitrap)中的运动,或在傅里叶转换离子回旋共振质谱仪的磁场中,测量离子质荷比。该仪器记录每个离子的相对丰度,其用来确定初始样品的化学、分子和/或同位素组成。在一些实施方案中,使用飞行时间型仪器(time-of-flight instrument),电场用来加速离子通过相同电位,并测量每个离子到达检测器所花费的时间。该方法依赖于每个离子的电荷是均匀的,使得每个离子的动能将是相同的。在该情况下影响速率的唯一变量是质量,其中较轻离子以更高的速度运行,并因此更快地到达探测器。获得的数据表示在质谱或直方图(强度相比于质荷比)中,其中峰代表离子化的化合物或片段。
为了获得蛋白样品的质谱数据,将样品上样至仪器上并电离化。电离可以通过,例如,电喷雾电离和基质辅助激光解吸/电离("MALDI")而进行。参见,例如Zenobi, "Ion Formation in MALDI Mass Spectrometry", 17 Mass Spectrometry Review, 337 (1998)。蛋白表征可以两种方式(自上向下或自下向上)之一来进行。自上向下方法涉及电离整个蛋白或较大的蛋白片段。参见,例如Allison Doerr, "Top-down Mass Spectrometry", 5 Nature Methods, 24 (2008)。自下向上方法涉及用蛋白酶将蛋白酶促或化学消化成组成肽。参见Biran Chait, "Mass Spectrometry: Bottom-Up or Top-Down?", 6 Science 65 (2006)。将获得的肽引入到仪器中,且最终通过肽质量指纹识别或串联质谱法进行确定。
在一些实施方案中,质谱分析可以与色谱分级(例如,液相色谱)组合。
可用于本发明的质谱数据可以通过肽质量指纹识别来获得。肽质量指纹识别包括将从通过蛋白水解消化生成的肽混合物的谱观察到的质量输入到数据库中,并且将观察到的质量与从已知蛋白在计算机芯片上的消化产生的片段的预测质量相关联。对应于样品质量的已知质量提供了已知蛋白存在于测试样品中的证据。
质谱数据可以通过串联质谱来获得。在一些实施方案中,串联质谱法通常利用碰撞诱导的解离,其导致肽离子与空气碰撞并成为片段(例如,由于碰撞所赋予的振动能量)。片段化过程产生在沿蛋白的各个位点的肽键处断裂的切割产物。对于许多给定肽序列之一,观察到的片段质量可以与预测质量的数据库进行匹配,并且可以预测蛋白的存在。参见,例如Eng, 5 An Approach to Correlate Tandem Mass Spectral Data of Peptides with Amino Acid Sequences in a Protein Database, JASMS, 976 (1994)。
在另一个实施方案中,串联质谱通过高能量碰撞诱导的解离(HCD)来进行,其在一些质谱仪上显示比碰撞诱导的解离与肽末端更近的片段产物离子。参见Olsen JV, Macek B, Lange O, Makarov A, Horning S, Mann M. Higher-energy C-trap dissociation for peptide modification analysis. Nat. Methods. 2007 Sep;4(9):709-12. Epub 2007 Aug 26. PubMed PMID: 17721543。
在另一个实施方案中,串联质谱法通过电子转移解离(ETD)进行,其基于离子-离子反应,其中不同的试剂化学离子将自由基供给肽离子,然后后者迅速片断化以形成产物离子。参见Mikesh LM, Ueberheide B, Chi A, Coon JJ, Syka JE, Shabanowitz J, Hunt DF. The utility of ETD mass spectrometry in proteomic analysis. Biochim Biophys Acta. 2006 Dec;1764(12):1811-22. Epub 2006 Oct 30. 综述. PubMed PMID: 17118725; PubMed Central PMCID: PMC1853258。某些片段化方法,诸如ETD,特别好地适合于“自上向下”蛋白质组学策略。其他片段化机制对于某些电离机制是特异性的,例如,诸如源后衰变(post-source decay,PSD)与基质辅助激光解吸电离(MALDI)是相容的,也是非常适合于“自上向下”蛋白质组学策略。
遗传物质数据库
根据本发明,将从开始多克隆免疫球蛋白群体观察到的质谱信息与源自遗传物质数据库的预测质谱信息相关联,以便从开始多克隆免疫球蛋白群体获得免疫球蛋白的免疫球蛋白链(或其可变区)的氨基酸(和基础核苷酸)序列。
如本文所使用的,遗传物质数据库包括编码多个免疫球蛋白链(或其可变区)的核酸序列。因此,可以从这种遗传物质数据库获得或源自这种遗传物质数据库的信息包括,例如,每种核酸分子的核苷酸序列信息,每种核酸分子的长度(以核苷酸为单位),由每种核酸分子编码的多肽或肽的氨基酸序列信息,由每种核酸分子编码的多肽或肽的质量,由每种核酸分子编码的多肽或肽的长度(以氨基酸为单位),由每种核酸分子编码的多肽或肽的质谱信息(例如,基于多肽或肽的氨基酸序列的预测质谱信息),和由每种核酸分子编码的多肽或肽的氨基酸序列。
在本发明的一些实施方案中,遗传物质数据库含有编码全长免疫球蛋白链(而不是仅仅其可变区)的核酸序列的遗传信息。在一些实施方案中,核酸序列由衍生所述序列的细胞表达(例如,转录成RNA和/或翻译成蛋白)。在具体实施方案中,遗传物质数据库包括编码动物的多种免疫球蛋白的免疫球蛋白链可变区的表达的核酸序列。在一些实施方案中,遗传物质数据库含有至少100种不同的表达的核酸序列。在其他实施方案中,遗传物质数据库含有至少1000种不同的表达的核酸序列。
编码免疫球蛋白链(或其可变区)的核酸分子可容易从含有B淋巴细胞的细胞群体(例如,外周血白细胞)获得。在一些实施方案中,核酸分子从脾细胞或单核细胞,诸如外周血单核细胞(PBMCs)获得。 在一些实施方案中,B淋巴细胞来自幼稚动物(例如,尚未暴露于寻求针对其的抗原特异性抗体的抗原的动物)。在一些实施方案中,幼稚动物已经暴露于非常少抗原(例如,无菌或无病原体环境中饲养的动物)。在一些实施方案中,幼稚动物是已经暴露于一般抗原、但尚未暴露于选择抗体的典型动物。
在一些实施方案中,由其获得编码免疫球蛋白链(或其可变区)的核酸序列的动物是以前已经暴露于抗原的动物。例如,动物可以是用抗原(例如,与佐剂混合的抗原,或与免疫原性载体诸如匙孔血蓝蛋白(KLH)偶联的抗原)免疫的动物,可以是感染包含抗原的病原体的动物(例如,当选择的抗原是HIV p24抗原时,感染HIV病毒的动物),或者可以是以前暴露于抗原。在一些实施方案中,动物是鸟(例如,鸡或火鸡)或哺乳动物,诸如灵长类动物(例如,人或黑猩猩),啮齿类动物(例如,小鼠、仓鼠、或大鼠),兔类动物(例如,兔或野兔),骆驼(例如,骆驼或美洲驼),或驯养动物诸如伴侣动物(例如,猫、狗或马),或家畜动物(例如,山羊、绵羊、或牛)。
应当理解,本发明的各个方面和实施方案的核酸序列不必来自单一动物。例如,本发明的各个实施方案中的一些核酸序列可能来自以前暴露于抗原的动物,且一些核酸序列可能来自幼稚动物。在本发明的一些实施方案中,核酸序列来自单一物种的动物。例如,当存在由其获得核酸序列的多种动物时,所有那些动物可以是相同的物种(例如,所有都是兔或所有都是人)。在一些实施方案中,核酸序列从单一物种的动物获得。在其他实施方案中,可以获得来自多于一个物种的动物的核酸序列。例如,核酸序列可以从小鼠和大鼠获得,并且基于这些序列的预测质谱可用于与多克隆抗体的肽片段的实际质谱信息相关联和/或比较,以生成特异性结合抗原的免疫球蛋白(或其可变区、抗原结合结构域或链)。在一些实施方案中,核酸序列从单一性别(例如,所有动物都是雌性)的动物获得。
由其收集多克隆抗体的动物和由其收集核酸序列的动物可以是相同的动物,或相同物种的动物,或同系动物(例如,两者都是Balb/c小鼠),或来自相同性别的动物(例如,两者都是雌性动物)。因此,来自多克隆抗体的抗原结合组分的MS2谱可以与源自从动物获得的核酸序列的理论MS2谱相关联,以鉴定编码抗原结合抗体的核酸序列。
也应当理解,核酸序列和多克隆抗体可以从动物细胞收集,其中细胞在从动物取出之后在体外培养,随后收集多克隆抗体(例如,从培养细胞的上清液或经培养的培养基)和从细胞收获核酸序列。该培养步骤可用于,例如,与其他血液或组织细胞相比扩增或富集B淋巴细胞(例如,以与红血细胞或上皮细胞相比富集B淋巴细胞)。本发明的各个实施方案中用于生成理论质谱的个别核酸序列的数目是无限的。例如,可以获得五或十或五十,或一百,或一千,或一百万,或十亿,或一万亿或更多种不同的核酸序列,且用于生成理论质谱。核酸序列可以来自任何来源,且可以来自来源的组合。例如,核酸序列可以通过对如本文所述的编码免疫球蛋白链可变区(或整个全长免疫球蛋白链,包括可变区和恒定区)的表达的核酸分子进行测序而获得。核酸序列也可以从基因组DNA获得,所述基因组DNA可能已经发生或尚未发生全V(D)J重组。核酸序列也可以从公开获得的来源获得。例如,来自许多动物物种的免疫球蛋白链可变区(和编码其的多核苷酸)的许多氨基酸序列(和核苷酸序列)是已知的(参见,例如,以下美国和PCT专利出版物(包括公布的美国专利) ,其中每一篇整体通过引用并入本文:US 20100086538; WO 2010/097435; US 20100104573; US 7,887,805; US 7,887,801; US 7,846,691; US 7,833,755; US 7,829,092。
由其获得核酸序列的B淋巴细胞可以来自任何血液或组织来源,包括但不限于,骨髓、胎儿血液、胎肝、炎症部位(例如,类风湿患者的关节滑液周围的发炎关节)、肿瘤(例如,肿瘤浸润性淋巴细胞)、外周血,在淋巴结中,在Peyer氏斑中,在扁桃体中,和在脾中或在任何淋巴器官中。在一些实施方案中,可以将整个组织(例如骨髓或淋巴结)处理(例如,彼此分离细胞并裂解),将遗传物质取出,并且将编码免疫球蛋白链(或其可变区)的核酸分子测序。
在一些实施方案中,B淋巴细胞从含有它们的组织或细胞群体(例如,外周血)富集,然后从B淋巴细胞分离遗传物质。根据本发明的各个实施方案中,用于从动物富集B淋巴细胞的方法是众所周知的。B淋巴细胞可以存在于机体的许多器官和区域,包括但不限于,骨髓、胎儿血液、胎肝、炎症部位(例如,类风湿患者的关节滑液周围的发炎关节)、肿瘤(例如,肿瘤浸润性淋巴细胞)、外周血,在淋巴结中,和在脾中。从这些组织样品(例如动物的外周血或脾)中,可以根据标准方法(例如,根据制造商的说明使用由 GE Healthcare, Piscataway, NJ商售的Ficoll-Paque PLUS或Ficoll-Paque PREMIUM试剂)分离白血细胞。然后,B淋巴细胞本身可以使用例如在B淋巴细胞上发现的细胞表面标志物进一步与其他白血细胞分离。B淋巴细胞细胞表面标志物包括但不限于细胞表面表达的免疫球蛋白链(例如,λ轻链,κ轻链和重链,诸如IgM或IgG)。额外的B淋巴细胞细胞表面标志物包括但不限于,CD21、CD27、CD138、CD20、CD19、CD22、CD72和CD79A。然而,额外的B淋巴细胞细胞表面标志物包括但不限于,CD38、CD78、CD80、CD83、DPP4、FCER2、IL2RA、TNFRSF8、CD24、CD37、CD40、CD74、CD79B、CR2、IL1R2、ITGA2、ITGA3、MS4A1、ST6GAL1、CD1C、CD138和CHST10。
这些B淋巴细胞表面标志物可以用于相继富集B淋巴细胞。例如,针对B淋巴细胞细胞表面标志物(例如,CD19)特异性的抗体可以偶联到磁珠(例如,由Invitrogen Corp., Carlsbad, CA商售的Dynabeads),和粘附至珠粒的与非CD19表达细胞分离的细胞(例如,CD19阳性细胞)。B淋巴细胞可以通过例如流式细胞术分选在其细胞表面表达免疫球蛋白链的细胞而从CD19阳性细胞进一步富集。因此,这些富集的B淋巴细胞可以进行分离以用于本发明的各个实施方案的方法中。
抗原特异性B淋巴细胞也可以使用作为诱饵的所需抗原进行直接纯化,以分离表达抗原特异性B细胞受体(膜免疫球蛋白)的B细胞。例如,B细胞可以添加至粘附所需抗原的柱上。抗原特异性B细胞将比非特异性B细胞或其他细胞(例如,红血细胞,巨噬细胞等)更慢地流过该柱。因此,可以使用这种方法富集抗原特异性B细胞。
来自动物的富集或未富集的B淋巴细胞(例如,通过各种方法富集)也可以进行体外细胞培养1或2或3或4或更多天,随后提取核酸。这种体外培养可以扩增B淋巴细胞的数量,因此与非B淋巴细胞相比富集它们。在一个非限制性实例中,CD27分离的人B淋巴细胞可以在提取核酸之前用各种细胞因子和细胞外分子混合物(诸如但不限于活化的T细胞条件培养基,或B细胞生长和/或分化因子的任何组合)处理,以便在从B淋巴细胞提取核酸之前刺激B淋巴细胞的生长和/或分化。其他生物分子也可以在体外培养过程中添加到组织培养基,以帮助生长、分化和/或体外免疫、和/或以上的任何组合。
可以使用标准方法(例如,苯酚:氯仿萃取;参见Ausubel等人,同上)从这些分离的、富集的或者刺激的B淋巴细胞提取核酸序列(例如,基因组DNA、hnRNA、mRNA等)。然后该核酸可以使用各种测序方法进行测序分析。
在一些实施方案中,核酸序列可以从生物材料直接测序(即,在测序前不经扩增)。用于从核酸序列直接测序的服务和试剂是商售的,例如,从Helicos BioSicences Corp. (Cambridge, MA)商售。例如,Helicos' True Single Molecule Sequencing可以直接测序DNA、cDNA和RNA。还参见美国专利号7,645,596; 7,037,687, 7,169,560;和出版物Harris等人, Science 320: 106-109, 2008; Bowers等人, Nat. Methods 6: 493-494, 2009; 和Thompson和Milos, Genome Biology 12: 217, 2011 (其中所有专利和出版物整体通过引用并入本文)。
在其他实施方案中,在获得序列信息之前扩增核酸序列(例如,通过聚合酶链式反应)。
在一个非限制性实例中,寡聚dT PCR引物用于RT-PCR中。在另一个非限制性实例中,使用本文所述PCR引物进行基因特异性RT-PCR,所述PCR引物诸如454特异性融合小鼠引物,454兔免疫球蛋白链融合引物或可变重和可变轻区域引物。在另一个实例中,针对小鼠重链和轻链群体的PCR引物具有PCT公开号WO2010/097435(通过引用并入本文)中所述的序列。
在有或没有B细胞富集的情况下,可以根据标准程序(参见,例如,Ausubel等人,同上)将纯化的遗传物质(DNA或mRNA)进行扩增(例如,通过PCR或RT-PCR),以便在NGS测序之前制备文库。
通过各种方式分离的上述B淋巴细胞也可以通过使用本领域中的方法诸如油乳剂封装或通过商用仪器诸如RainDance技术(RainDance Technologies, Inc., Lexington, MA)进行单细胞封装。然后这些封装的B淋巴细胞可以与适当的单细胞RT-PCR试剂(例如,由Qiagen作为目录号210210销售的试剂)以及适当的扩增引物融合,以便从每个单个B细胞生成连接的重链和轻链PCR产物。连接或重叠PCR是本领域中已知的,且对于将2个DNA片段缝合为一的各种分子生物学应用是常规实践的(对于重叠PCR,参见例如Meijer P.J.等人, J. Mol. Biol. 358(3):764-72, 2006)。该方法允许测序过程中保存和鉴定同源配对。
DNA测序方法
本领域中众所周知的且通常可用的DNA测序方法可用于获得本发明的各个实施方案中的核酸序列。该方法可以采用这样的酶如DNA聚合酶I的Klenow片断、SEQUENASE? (US Biochemical Corp, Cleveland, Ohio)、Taq聚合酶(Invitrogen)、热稳定T7聚合酶(Amersham, Chicago, Ill.)、DNA连接酶(例如,来自T4)或重组聚合酶和校正外切核酸酶的组合诸如由Gibco BRL (Gaithersburg, Md.)出售的ELONGASE扩增系统。该过程可以借助于仪器自动进行,诸如 Hamilton Micro Lab 2200 (Hamilton, Reno, Nev.)、Peltier Thermal Cycler (PTC200; MJ Research, Watertown, Mass.)以及ABI 377 DNA测序仪(Applied Biosystems)。
对核酸分子测序且因此生成本发明的各个实施方案的核酸序列(例如,以填充遗传物质数据库)的非限制性方法包括Sanger方法(参见,例如,Sanger等人,Nature 24:687-695, 1977),Maxam-Gilbert方法(参见,例如,Maxam和Gilbert, Proc. Natl. Acad. Sci. USA 74:560-564, 1977)和焦磷酸测序(参见,例如,Ronaghi等人, Science 281 (5375):363, 1998和Ronaghi等人, Analytical Biochemistry 242 (1):84, 1996)。可以用来获得多核苷酸序列的另一种非限制性测序方法焦磷酸测序使用荧光素酶来生成用于检测添加到新生DNA的单个核苷酸(dATP、dTTP、dGTP、或dCTP,统称“dNTPs”)的光,并且组合的数据被用于生成序列读出值。
在一些实施方案中,核酸序列使用深度测序或下一代测序获得。常规DNA测序中的一个限速步骤由需要通过凝胶电泳分开随机终止的DNA聚合物而导致。下一代测序设备绕过该限制,例如,通过在固体表面上物理排列DNA分子且在原位确定DNA序列,而不需要凝胶分离。这些高通量测序技术允许平行测序许多核酸分子。
因此,数千或数百万的不同核酸分子可以同时测序(参见Church, G.M., Sci. Am. 294 (1): 46–54, 2006; Hall, N., J. Exp. Biol. 210(Pt. 9): 1518-1525, 2007; Schuster等人, Nature Methods 5(1): 16-18, 2008; 和MacLean等人, Nature Reviews Microbiology 7: 287-296, 2009)。存在用于进行下一代测序的各种不同方法和机器,其中任一种可以用来生成核酸序列。对于许多下一代测序技术的概述,参见Lin等人, Recent Patents on Biomedical Engineering 1:60-67, 2008。
例如,Shendure, J.等人, Science 309(5741):1728-32, 2005和美国专利公开号20070087362描述了使用基于连接的测序方法的polony下一代测序方法(还参见美国专利号5750341)。由Applied Biosystems (a LifeTechnolgies Corp. company, Carlsbad, CA)商售的SOLiD技术采用边连接边测序(sequencing by ligation)。使用SOLiD技术,待测序的DNA片段的文库通过乳液PCR扩增,并且在文库中的多个片段中,单一片段种类将结合至单一磁珠(所谓的克隆珠粒)。连接到磁珠的片段将具有连接的通用的P1接头序列,使得每个片段的开始序列是已知的和相同的。然后选择与文库模板内的P1适配子序列杂交的引物。一组四个荧光标记的二碱基探针竞争与测序引物连接。二碱基探针的特异性通过查询各连接反应中的每个第一和第二碱基而实现。
Margulies等人, Nature 437:376-380, 2005和美国专利号7,211,390; 7,244,559; 和7,264,929的另一种下一代测序方法描述了焦磷酸测序法的平行版本,其扩增油溶液中的水滴内的DNA(乳液PCR),每个液滴含有连接到单一引物包被珠粒的单一DNA模板。使用测序仪(由454 Life Sciences, a Roche company, Branford, CT商售的Genome Sequencer FLX System机器),将寡核苷酸适配子连接至片段化的核酸分子,然后固定至微观珠粒的表面,然后在油滴乳液中PCR扩增。然后珠粒在多皮升体积孔中分离,各孔含有单一珠粒、测序酶和dNTP。dNTP掺入互补链释放焦磷酸,其产生ATP,所述ATP进而生成光,所述光然后可以作为分析图像进行记录。
美国专利号7,115,400描述了用于固相扩增核酸分子的另一种技术。这允许大量不同的核酸序列进行阵列且同时进行扩增。该技术在由Solexa (Illumina, Inc.)商售的Genome Analyzer系统中实施。该技术中,DNA分子首先连接至载片上的引物,并且扩增,使得形成局部克隆集落(桥扩增)。添加四种类型的ddNTPs,且洗走非掺入的核苷酸。与焦磷酸测序不同,该DNA只可以一次延伸一个核苷酸。相机拍摄荧光标记的核苷酸的图像,然后染料连同末端3'受体阻断剂通过化学方法从DNA去除,以允许下一个循环。
编码免疫球蛋白链可变区的多核苷酸序列可以使用部分核苷酸序列加以延伸并采用本领域已知的各种方法来检测上游序列如启动子和调节元件。例如,可以采用的一种方法,“限制位点”PCR,使用通用引物来恢复(retrieve)相邻于已知基因座的未知序列 (Sarkar, G., PCR Methods Applic. 2: 318-322 (1993))。尤其是,在存在接头序列的引物和对于已知区特异性的引物的情况下首先扩增基因组DNA。示例性的引物是那些在本文的实施例4中描述的引物。扩增序列然后经受第二轮PCR,其中使用相同的接头引物和第一种引物内部的另一种特异性引物。借助于适当的RNA聚合酶转录每轮PCR的产物并利用逆转录酶进行测序。
利用基于已知区的趋异性引物,反向PCR还可以用来扩增或延伸序列(Triglia等人, Nucleic Acids Res. 16: 8186 (1988))。利用OLIGO 4.06引物分析软件(National Biosciences Inc., Plymouth, Minn.)、或另一种适当的程序,引物可以被设计成长度为22-30个核苷酸,具有50%或更大的GC含量,以及在约68-72℃的温度下退火至靶序列。该方法使用多种限制酶以在基因的已知区域中产生适当的片断。然后通过分子内连接反应环化该片断并用作PCR模板。
可以使用的另一种方法是捕获PCR,其涉及DNA片断的PCR扩增,其中DNA片断相邻于人和酵母人工染色体DNA中的已知序列 (Lagerstrom等人, PCR Methods Applic. 1: 111-119 (1991))。在该方法中,多限制酶消化和连接作用还可以用来在进行PCR以前将工程改造的双链序列放入DNA分子的未知部分中。可以用来恢复未知序列的另一种方法是描述在Parker等人, Nucleic Acids Res. 19: 3055-3060 (1991))中的方法。另外,可以使用PCR、套式引物、以及PROMOTERFINDER?文库来步查(walk in)基因组DNA(Clontech, Palo Alto, Calif.)。这种方法避免需要筛选文库并且可用于发现内含子/外显子接头。
应当理解,在测序之前,可以针对编码免疫球蛋白的那些核酸分子进一步筛选来自B淋巴细胞的核酸。为了做到这一点,可以采用对于编码免疫球蛋白的核酸分子特异性(或对于与之邻近的区域特异性)的引物。
如本文所使用的,“引物”是指一种核酸序列,其可以是至少约15个核苷酸,或至少约20个核苷酸,或至少约30个核苷酸,或至少约40个核苷酸长度。对于特定核酸分子特异性的引物是指包括在PCR退火条件下(例如,60℃持续30秒)与核酸分子的一部分杂交的引物。在一些实施方案中,对于特定核酸分子特异性的引物是与该核酸分子互补的引物。
用于对核酸序列测序的引物可被称为测序引物。用于通过聚合酶链式反应(PCR)对靶核酸序列扩增的引物也可以被称为PCR引物或扩增引物(参见例如Sambrook等人,同上和Ausubel等人,同上中PCR的描述),其全部公开内容在此通过引用并入本文。
在根据本发明的各个实施方案获得核酸序列的一个非限制性实例中,来自B淋巴细胞的总核酸可以是释放的单链(例如,通过将核酸加热到94-98℃下持续至少一分钟。然后,可以将单链核酸通过已粘附单链引物的固体支持物(例如,柱或凝胶),所述单链引物对于编码免疫球蛋白的核酸分子的非变体区域或与之邻近的非编码区(例如,免疫球蛋白基因启动子、增强子和/或内含子)是特异性的。免疫球蛋白的这些非变体区域的一些非限制性实例包括重链恒定区、和轻链恒定区、和重链或轻链的FR1区。允许核酸杂交至固相支持物结合的引物,并且去除未杂交的核酸。去除之后,杂交核酸(针对编码免疫球蛋白的核酸分子对其富集)从引物释放,通过例如,加热,或增加EDTA在缓冲液中的浓度。
在本发明的另一个实施方案中,无论是否针对编码免疫球蛋白的核酸分子富集来自B淋巴细胞的核酸,编码免疫球蛋白的核酸分子可以进行扩增以增加其拷贝数。该扩增可以进行,例如,通过使用对于编码免疫球蛋白的核酸分子的非变体区域或与其邻近的非编码区特异性的引物的PCR扩增。
在所有上面用于根据本发明的各个实施方案获得核酸序列的方法中,将理解,用于生成编码免疫球蛋白链可变区的核酸序列的引物(例如,测序或PCR引物)可以是通用的(例如,聚A尾部),或对于编码免疫球蛋白的序列可以是特异性的。
在一些实施方案中,由其获得编码免疫球蛋白基因的核酸序列的起始材料是基因组DNA。例如,如果免疫球蛋白链可变区来自人,则可以选择引物(例如,测序引物和/或PCR引物)与免疫球蛋白链基因启动子是相同的,或与其杂交。例如,人基因组序列是已知的。由于重链编码基因存在于染色体14上且轻链编码基因存在于染色体22(λ轻链)和2(κ轻链)上,所以设计与重链编码基因和轻链编码基因的调控元件杂交的引物对于普通熟练的生物学家而言是常规的。这些调控元件包括,但不限于,启动子、增强子和内含子。
免疫球蛋白可变区特异性引物对于小鼠免疫球蛋白同样可以很容易地确定,因为已知鼠κ轻链基因位于染色体6上,且已知鼠重链基因位于染色体12上。
在另一个非限制性实施方案中,由其获得编码免疫球蛋白基因的核酸序列的起始材料是mRNA或由mRNA反向翻译的cDNA。在该实例中,为了获得编码免疫球蛋白可变区的核酸序列,可以选择引物与mRNA或mRNA的对应cDNA的互补的富含TTTT(SEQ ID NO:306)的序列的聚A尾部是相同的,或与其杂交。可替代地,或额外地,也可以选择引物与编码FR1的核酸序列是相同的或与其杂交。可替代地,或额外地,也可以选择引物与编码CH区域之一(即,CH1、CH2、或CH3)和/或VH区的核酸序列的部分(或全部)是相同的或与其杂交。
测序错误可以由于使用通用简并引物来对编码来自杂交瘤的免疫球蛋白的核酸分子进行测序而导致。例如,Essono等人,Protein Engineering, Design and Selection, pp.1-8, 2009描述了组合测序与对应Ig链的肽质谱指纹识别以确定由杂交瘤克隆产生的单克隆抗体的正确序列的方法。然而,在本发明的各个实施方案的非限制性方法中,测序错误的存在将仅仅增加不同核酸序列的数目。不同于Essono等人,同上,由于本发明的各个实施方案的方法允许从起始多克隆抗体群体(其中生成的抗体实际上可能不存在于起始多克隆抗体群体内)生成单一抗体(重和轻链两者或其可变区),遗传物质数据库中具有大量序列(观察到的肽数据库的质谱数据与其相关联)是一个优点。
来自遗传物质数据库的预期质谱信息
根据本发明的各个实施方案,一旦生成核酸分子的核苷酸序列,可以仅基于核苷酸序列信息生成额外信息。例如,核苷酸序列信息可以使用遗传代码翻译成预期的氨基酸序列。尽管普通技术人员可以容易地使用遗传代码将核苷酸序列翻译成氨基酸序列,但是可以使用几种自动翻译工具(这是可公开获得的),诸如来自Swiss Institute of Bioinformatics的ExPASy翻译工具或来自EMBL-EBI的EMBOSS Transeq翻译工具。
类似地,由核酸序列编码的预测氨基酸序列的预测质谱信息可以由普通技术人员容易地确定。例如,虚拟(即在计算机芯片上)消化由核酸序列编码的预测多肽之后,肽片段的预测质谱可以通过使用以下生成:标准的可公开获得的软件算法工具,包括但不限于Sequest 软件(来自Thermo Fisher Scientific, Inc., West Palm Beach, FL)、the Sequest 3G软件(来自Sage-N Research, Inc., Milpitas, CA)、the Mascot软件(来自Matrix Science, Inc., Boston, MA; 还参见Electrophoresis, 20(18) 3551-67 (1999))、和the X!Tandem软件(来自The Global Proteome Machine Organization的开放源,其用途描述于Baerenfaller K.等人, Science 320:938-41, 2008)。
如本文所使用的,词语“预测的”、“理论的”和“虚拟的”可以互换用于指核苷酸序列、氨基酸序列或质谱,所述核苷酸序列、氨基酸序列或质谱源自在计算机芯片上(即,在计算机上)转录和/或翻译(对于预测的核苷酸序列和氨基酸序列)或在计算机芯片上消化和/或质谱分析(对于预测的质谱)来自核酸序列的信息。例如,核酸序列源自从如本文所述的B淋巴细胞获得的基因组核酸分子。计算机芯片上翻译基因组DNA之后预测例如源自基因组DNA的mRNA的核苷酸序列。该预测的mRNA(或cDNA)然后可以在计算机芯片上翻译,以产生预测的氨基酸序列。然后可以将预测的氨基酸序列在计算机芯片上用蛋白酶(例如胰蛋白酶)和/或化学蛋白切割试剂(例如溴化氰)消化以产生预测的(或理论的或虚拟的)肽片段。然后可以将该虚拟的肽片段在计算机芯片上分析以产生预测质谱信息。因此,预测的质谱信息、预测的肽片段、预测的氨基酸序列和预测的mRNA或cDNA序列都可以源自从B淋巴细胞(例如,来自动物)收集的核酸序列。
在某些实施方案中,用于将预测的多肽消化生成预测的肽片段和最终预测质谱的一种或多种蛋白酶和/或化学试剂与如上所述用于消化多克隆抗体的起始群体的一种或多种蛋白酶和/或一种或多种试剂是相同的。
将观察的质谱与预测的质谱相关联
如上所述,源自起始多克隆抗体群体的片段通过质谱仪生成许多观察的质谱。鉴于多克隆群体内可能存在大量不同的免疫球蛋白(各自具有不同的氨基酸序列,其用质谱仪分析),获得的观察到的质谱将难以装配回功能性免疫球蛋白链可变区。在本发明的各个实施方案的方法中,因为编码核酸序列是可用的,所以不必装配观察到的质谱数据。相反,将观察到的质谱与源自遗传物质数据库的核酸序列的预测的质谱相关联,以便从开始多克隆免疫球蛋白群体获得特异性结合抗原的免疫球蛋白的全长免疫球蛋白链(或其可变区)的氨基酸(和基础核苷酸)序列。
同样如上所述,遗传物质数据库可以源自从免疫动物的B细胞所有组成成分分离的核酸分子,包括编码全长免疫球蛋白重链和轻链和其可变区的核酸分子。仅仅基于来自遗传物质数据库的信息(例如,可变区序列的频率排序)鉴定编码抗原特异性免疫球蛋白的核酸的尝试可能会错过以低频存在但表现出优异抗原特异性活性的那些免疫球蛋白。然而,根据本发明的各个实施方案,通过将来自遗传物质数据库的预测的质谱信息与如本文公开的来自实际循环多克隆抗体的观察到的质谱信息相关联,可以选择对应于循环多克隆抗体内的免疫球蛋白的遗传物质数据库中的那些免疫球蛋白链(或其可变区)。
“关联”是指,将源自起始多克隆抗体的观察到的质谱信息和源自遗传物质数据库的预测的质谱信息彼此交叉参考和比较,从而使得可以从遗传物质数据库鉴定或选择对应于起始多克隆群体中抗原特异性免疫球蛋白的免疫球蛋白重链和/或轻链(或其可变区)的免疫球蛋白重链和/或轻链(或其可变区)。
在具体实施方案中,关联过程包括比较观察到的质谱信息和预测的谱信息来鉴定匹配。例如,每个观察到的谱可以针对源自遗传物质数据库的预测质谱的集合进行搜索,每个预测的谱与来自遗传物质数据库的肽序列可标识地相关。一旦发现匹配,即,观察到的质谱与预测的质谱相匹配,因为每个预测的质谱与遗传物质数据库中的肽序列可标识地相关,所以观察到的质谱被称为已经发现其匹配的肽序列 – 这种匹配在本文中也称为“肽谱匹配”或“PSM”。因为存在大量谱需要进行搜索和匹配,所以该搜索和匹配过程可以通过计算机执行的功能和软件,诸如SEQUEST算法(Sage-N Research, Inc., Milpitas, CA)来执行。
在一些实施方案中,搜索与匹配涉及免疫球蛋白的功能结构域或片段,诸如可变区序列、恒定区序列,和/或一种或多种CDR序列。例如,观察到的谱只针对源自免疫球蛋白的V区(和/或CDR3序列)的预测质谱进行搜索,以鉴定V-区(和/或CDR3) PSMs。在其他实施方案中,搜索与匹配涉及全免疫球蛋白重链或轻链序列。
已经完成搜索和匹配后,对遗传物质数据库中的免疫球蛋白重链或轻链分析,并基于以下参数中的一个或多个进行选择: 独特肽的数量、谱共享、氨基酸序列覆盖率、肽的计数(总肽计数或独特肽计数)、编码核酸序列的频率和克隆相关性。
涉及序列或区域(例如,重或轻链序列、V-区序列或CDR序列)的术语“覆盖率”被定义为映射到该序列或区域且具有匹配的观察到的谱的肽中已经鉴定的序列内的氨基酸总数除以该序列或区域中氨基酸的数量。覆盖率越高,该序列或区域在实际多克隆群体中可能出现越多。
“独特肽的数量”是指映射到单一蛋白序列(例如,单一免疫球蛋白重链或轻链或其可变区)的观察到的独特肽的数量。数量越高,免疫球蛋白链在多克隆群体中可能存在越多。在具体实施方案中,基于免疫球蛋白链或其可变区中至少5、6、7、8、9、10、11、12或更多的独特肽的数量进行免疫球蛋白链的选择。
“谱共享”通过映射到序列的肽的总数除以映射到整个遗传数据库的置信PSMs的总数而确定。谱共享提供了肽的人可读计数,其表示为映射到特定V区序列的PSMs的百分比。
涉及蛋白序列(例如,CDR3区或可变区)的术语“肽计数”是指从观测到的质谱鉴定出匹配蛋白序列的肽的次数。例如,CDR3区的计数是指从观测到的质谱鉴定出匹配CDR3区的肽的次数。可变区的计数是指从观测到的质谱鉴定出匹配可变区的肽的次数。涉及蛋白序列的“总肽计数”是指从观察到的质谱鉴定出匹配蛋白序列的任何肽(独特或非独特)的次数。“独特肽计数”是指从观察到的质谱鉴定出匹配蛋白序列的独特肽的次数。如果已经多次从观测到的质谱鉴定出相同肽,则在确定总肽计数中将考虑观察到该肽的总次数,但对于确定独特肽计数,该肽将只计数一次。
在具体实施方案中,基于序列覆盖率选择免疫球蛋白重链或轻链。在其他实施方案中,基于序列覆盖率和一种或多种其他参数(包括独特肽的数量、谱共享、总肽计数、独特肽计数、编码核酸序列的频率或克隆相关性)的组合进行选择。
上面参数可以对于全长重或轻链或对于免疫球蛋白重链或轻链的一个或多个部分,例如,可变区和CDR(例如,CDR1、CDR2或CDR3,尤其是CDR3)独立地确定。在某些实施方案中,免疫球蛋白链(或其可变区)的选择基于V区覆盖率和/或CDR覆盖率(例如,CDR3覆盖率)来进行。
免疫球蛋白重链或轻链(或其可变区)的选择可以基于一种或多种参数的绝对值,或基于相关参数的绝对值的排序来进行。当考虑特定参数的排序时,选择排序最高的10、20、30、40、50、60、70、80、90、100或更多的序列,而不论该参数的绝对值。当考虑参数值(例如,在一些实施方案中,序列覆盖率的百分比)时,免疫球蛋白链的选择基于至少10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、98%或更高的CDR覆盖率(诸如CDR3覆盖率);额外地或可替代地,基于至少5%、10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%或更高的V区覆盖率来进行。
在一些实施方案中,进行系统发生分析来确定重链可变区、轻链可变区或一个或多个CDR(例如,CDRH3或CDRL3)的克隆关联性。重链和轻链的核酸序列与种系序列相比的变化或突变可以提供抗原暴露后抗体的亲和力成熟的证据。克隆相关性可以用来作为选择抗体序列中的一个因素。系统发生分析可以通过本领域中已知的方法进行,例如,描述于以下中的方法:Dereeper等人, 2008, Nucl. Acids Res., 36(Web Server issue):W456-459; Dereeper等人, 2010, BMC Evol. Biol., 10:8, 和在www.phylogeny.fr/version2_cgi/index.cgi在线获得。在一些实施方案中,整个重链或轻链可变区通过同源性分组,然后通过CDR(例如,CDR3)同源性进一步分组。
然后可以成对表达选择的重链和轻链序列以装配成单克隆抗体,然后分析所述单克隆抗体以确认抗原特异性。选择的重链和轻链序列的配对可以是完全随机的,也可以考虑上述的一个或多个参数,包括序列覆盖率、独特肽数量、谱共享、总肽计数、和独特的肽计数。
在一些实施方案中,具有特定肽序列的抗体群体的丰度可以使用重同位素标记(例如,AQUA)的肽来确定。参见例如,WO 03/016861和Gerber等人, 2003, 100:6940-45。这些方法采用将已知量的至少一种重同位素标记肽标准品(其具有可以通过LC-SRM层析法检测的独特特征)引入消化的生物样品中,以便通过与肽标准品的比较来确定生物样品中具有相同序列和蛋白质修饰的肽的绝对量。肽对于一种抗体可以是独特的,或者可以存在于多种(例如,克隆相关的)抗体中。在一些实施方案中,肽可以包括至少一部分的CDR(例如,CDR3)。抗体群体丰度的定量可用于监测血清抗体组成(例如,接种受试者后的血清抗体组成)的方法。
应当指出,其氨基酸序列(或核酸序列)使用本发明的各个实施方案的非限制性方法生成的特异性结合抗原的免疫球蛋白实际上并不需要存在于免疫球蛋白的起始多克隆群体内。相反,本发明的各个实施方案的非限制性方法简单地允许快速产生特异性结合抗原的免疫球蛋白,而无论该免疫球蛋白实际上是否存在于起始多克隆群体内。例如,生成的具有最高期望质量(例如,对于抗原或所需同种型(例如,IgG2a)具有最高结合亲和力(或最低KD))免疫球蛋白可能是由于来自多克隆群体中第一抗体的轻链与来自多克隆群体中第二抗体(即,不同于第一抗体)的重链装配在一起的结果。根据标准方法进一步表征获得的生成的免疫球蛋白(例如,对于抗原或同种型的结合亲和力)。
制备重组抗体的方法
一旦阐明特异性结合抗原的抗体的免疫球蛋白链(或其可变区)的核苷酸序列,可以生成包含该序列的核酸分子。
例如,如果由其获得编码免疫球蛋白链(或其可变区)的核酸分子的起始群体是cDNA文库,可以从该文库(例如,通过用与阐明序列的部分相同或能够杂交的引物筛选文库),或通过使用设计用于扩增阐明的核酸序列的引物从该文库PCR扩增核酸分子而容易地获得包含阐明序列的核酸分子。
可替代地(或此外),包含阐明的核苷酸序列的核酸分子可以通过使用标准的DNA合成机器简单地人工生成核酸分子而生成。许多DNA合成机器是商售的,包括但不限于从BioAutomation, Plano, TX获得的MerMade系列合成仪(例如,MerMade 4、Mermade 6、MerMade 384等);由Applied Biosystems (现在是Life Technologies, Corp., Carlsbad, CA的部分)商售的各种DNA/RNA合成仪。几家公司还提供DNA合成服务(例如,BioPioneer、Bio S&R, Biomatik、Epoch BioLabs等)。
表达编码免疫球蛋白的重链和轻链的核酸以产生重组免疫球蛋白的方法是已知的(参见,例如,美国专利公开号6,331,415;5,969,108;7,485,291;US 2011-0045534;和PCT公开号WO 2011/022077)。重组免疫球蛋白可以在各种细胞中制备,所述细胞包括,但不限于,昆虫细胞(例如,SF9细胞)、仓鼠细胞(例如,CHO细胞)、鼠细胞(例如,NIH-3T3细胞)、灵长类细胞(例如,COS细胞)、人细胞(例如,Hela细胞)、和原核细胞(例如大肠杆菌细胞)。在一些实施方案中,表达本发明的各个实施方案的重组免疫球蛋白的细胞能够以与由其最初衍生免疫球蛋白的物种类似的方式将二次修饰(例如,糖基化)添加到重组免疫球蛋白。例如,当其片段用来生成观察到的质谱数据的多克隆抗体群体是从人收集时,可以使用人细胞(或其与人细胞类似或相同地使蛋白糖基化的细胞)。
为了获得特异性结合抗原的重组免疫球蛋白(或其抗原结合片段)的核酸序列在细胞中的表达,可以将核酸序列连接到含有适当调控序列的载体(例如,质粒或逆转录病毒载体)中,从而使得插入的核酸序列在其中引入核酸序列的细胞中表达。这些调控序列包括,例如,启动子、增强子、内含子受体元件、聚腺苷酸化位点等。可以采用任何方法将重组免疫球蛋白的核酸序列(或含有其的载体)引入到细胞中,所述方法包括,但不限于,电穿孔,通过化学方法(例如,CaPO4、DEAE-葡聚糖、聚乙烯亚胺)转染、感染、转导、脂质体融合等(参见,例如,Ausubel等人,同上中的方法)。
根据本发明的一些实施方案,随机选择免疫球蛋白重链和免疫球蛋白轻链以装配成免疫球蛋白(或其可变区或抗体结合结构域)。例如,将使用来自多克隆抗体的肽片段的实际质谱与由核酸序列编码的预测肽的预测质谱的关联性,以获得包含肽片段的免疫球蛋白链的核苷酸序列或预测的氨基酸序列。然后在其中两条编码的免疫球蛋白链将装配成完整抗体的条件下将获得的免疫球蛋白链的核苷酸序列和/或与免疫球蛋白链的第二个类似地获得的核苷酸序列随机共表达,其中第二核苷酸序列编码完整抗体的另一条链。
用于共表达两个核苷酸序列(各自编码免疫球蛋白链)(例如,在细胞中)使得装配完整免疫球蛋白的条件是已知的(参见,例如,美国专利号5,969,108;6,331,415;7,498,024;7,485,291;和美国专利公开号20110045534,所有整体通过引用并入本文)。因为存在可以使用本文所述的方法获得的许多不同的核苷酸序列,本发明考虑使用机器人和高通量方法来筛选编码的免疫球蛋白来生成特异性结合抗原的免疫球蛋白。
如本文所使用的,“装配的”或“装配”是指,抗体(或其片段)的轻链和抗体(或其片段)的重链以其中两条链结合以生成抗体(或其片段)的方式组合在一起。在一些实施方案中,在装配的抗体(或其片段)中,来自重链和轻链两者的氨基酸残基贡献装配的抗体(或其片段)的抗原结合结构域。在一些实施方案中,装配的抗体(或其片段)包含与重链(或其片段)共价键合的轻链(或其片段)。在一些实施方案中,装配的抗体(或其片段)包含与重链(或其片段)非共价键合的轻链(或其片段)。
在一些实施方案中,在重组抗体装配前,通过重组分子生物学技术或基因合成技术合成上述基因组分析中鉴定的免疫球蛋白链(或其可变区)的核苷酸序列或氨基酸序列。例如,在装配前,可以在核苷酸或肽合成仪上合成核苷酸或氨基酸序列。或者,核苷酸或氨基酸序列可以通过以下重组表达:将核苷酸序列克隆到表达载体(例如,来自Invitrogen, Carlsbad, CA的pCDNA3.1)中,并且在用表达载体转染的细胞(例如,HeLa细胞、CHO细胞、COS细胞等)中表达编码的多肽。在一些实施方案中,装配步骤发生在转染的细胞中(例如,用一种或多种表达载体转染单一细胞,所述表达载体包含编码一条重链和一条轻链的核酸序列,其中重链和轻链将在转染的细胞中作为多肽表达)。
在本发明的各个实施方案中,重组抗体是分离的。如本文所使用的,“分离的”(或“纯化的”)是指抗体基本上不含其天然相关的其他生物材料,或不含其他生物材料,例如,不含源自已经遗传工程改造以表达本发明的抗体的细胞的其他生物材料。例如,分离的重组抗体是与宿主细胞中的其他组分(例如,内质网或胞质蛋白和RNA)物理上分离的抗体。同样地,从血液血清和/或血浆中纯化的抗体是与其他血清或血浆组分(例如白蛋白或细胞)相分离的抗体(使用,例如,抗体与蛋白A的粘附,其中非抗体血清组分不会粘附到蛋白A)。因此,本发明的分离的抗体(或分离的免疫球蛋白)包括至少70-100%纯的抗体,即,在其中该抗体构成总组合物的70-100重量%的组合物中存在的抗体。在一些实施方案中,本发明的分离的抗体是75重量%-99重量%纯,80重量%-99重量%纯,90重量%-99重量%纯,或95重量%至99重量%纯。本发明的各个非限制性实施方案的抗体纯度的相对程度由众所周知的方法容易地确定。
在一些实施方案中,重组抗体(或其可变区)在免疫测定中进一步筛选或分析,以验证该抗体特异性结合抗原。在一些实施方案中,免疫测定是标准免疫测定,诸如流式细胞术测定(例如,FACS扫描)、酶联免疫吸附测定(ELISA)、Western印迹测定、免疫组织化学测定、免疫荧光测定、放射免疫测定、中和测定、结合测定、亲和力测定、或蛋白或肽免疫沉淀测定。所有这些免疫测定是众所周知的标准测定,并且已经在标准方法书籍中很好地描述(参见,例如,Ausubel等人,同上; Coligan等人,同上; Harlow和Lane,同上)。
治疗性抗体
本发明的各个非限制性实施方案和方法可用于,例如,分离具有治疗价值的抗体。例如,在动物对病原体的正常免疫应答的过程中,对病原体抗原具有最高特异性的抗体可能需要数周来产生。这是因为产生抗体的B淋巴细胞首先必须由适当的T淋巴细胞刺激,所述适当的T淋巴细胞在由动物的每种有核细胞表达的主要组织相容性复合物的背景下还识别递呈在抗原递呈细胞上的抗原。最初对抗原应答的B淋巴细胞产生特异性结合抗原的抗体。然而,最高亲和力的抗体实际上是由B淋巴细胞产生的那些抗体,所述B淋巴细胞已经结合它们的抗原(通过与其他细胞表面抗原复合的细胞表面表达的免疫球蛋白以形成B细胞受体),并且在通过B细胞受体和其他细胞(包括T淋巴细胞)刺激后,经历亲和力成熟以产生对其特异性抗原具有高亲和力的抗体。如果动物应该再次遇到相同病原体,则已经经历亲和力成熟的这种B淋巴细胞(或其具有相同抗体特异性的后代)在动物中可用于快速产生高亲和力抗体。
T淋巴细胞和B淋巴细胞在见到该抗原的第一次(例如,动物被病原体感染的第一次)对抗原应答的这种紧密调节对于防止自身免疫或不当的免疫应答是必要的。然而,一个缺点是,到抗原特异性B淋巴细胞分泌对于抗原具有最高亲和力和特异性的抗体时,快速生长的病原体在动物体内可能已经生长到它不再容易清除的程度。在本发明的一些实施方案中,所述方法允许快速开发抗原特异性抗体,其跳过首先分离分泌抗体的抗原特异性B淋巴细胞和永生化该淋巴细胞的耗时过程。
因此,在另一个方面,本发明提供了包含重组抗体和药学可接受的载体的治疗组合物。
如本文中所用,“药学可接受的载体”包括:当与活性成分(例如,根据本发明的各个实施方案制备的重组抗体)组合时,使该成分保持生物活性且对受试者的免疫系统为非反应性的以及当递送时,对受试者是非毒性的任何物质。实例包括但不限于标准药物载体中的任一种,诸如磷酸盐缓冲盐水溶液、水、乳剂诸如油/水乳剂和多种类型的湿润剂。用于气溶胶或肠胃外施用的稀释剂的非限制性实例为磷酸盐缓冲盐水、生理(0.9%)盐水、林葛尔氏溶液(Ringer′s solution)及右旋糖溶液。溶液的pH值可从约5至约8,或从约7至约7.5。其他载体包括持续释放制剂,诸如含有抗体的固体疏水聚合物的半透性基质,此类基质系呈成形对象的形式,例如膜、脂质体或微粒。本领域技术人员应了解,根据例如施用途径及施用抗体的浓度,某些载体可为更优选的。通过众所周知的方法来配制包含此类载体的组合物(参见例如Remington's Pharmaceutical Sciences,第18版,A.Gennaro编,Mack Publishing Co., Easton, Pa., 1990;及Remington, The Science and Practice of Pharmacy, 第20版,Mack Publishing,2000)。
尽管也可以在本发明的药物组合物中采用本领域普通技术人员众所周知的任何合适的载体,但是载体的类型取决于施用方式而不同。在本发明的各个实施方案中,本文所述的非限制性药物组合物(例如,含有结合剂或编码结合剂的多核苷酸)的多种递送技术在本领域是众所周知的,如Rolland, 1998, Crit. Rev. Therap. Drug Carrier Systems 15:143-198和其中引用的参考文献所述的那些。
治疗方法
在另一个方面,本发明提供治疗患有或疑似患有特征在于疾病抗原的疾病的动物的方法,其中所述方法包括施用有效量的治疗组合物,所述治疗组合物包含根据本发明的各个实施方案的方法制备的特异性结合抗原的免疫球蛋白,其中由治疗组合物的免疫球蛋白特异性结合的抗原和疾病抗原是相同的。
在一些实施方案中,动物是人或驯养动物(例如狗、猫、牛、山羊、绵羊、鸡、火鸡、美洲驼、鸸鹋、大象或鸵鸟)。
如本文所使用的,关于疾病和指示的疾病抗原(例如,来自AIDS的HIVgp120抗原)的短语“特征在于…”是指其中指示的疾病抗原存在于患有该疾病的动物中的疾病。在一些实施方案中,疾病抗原由来自疾病的病因病原体(例如病毒)的核酸编码。在一些实施方案中,疾病抗原由动物的基因组编码(例如,由患有慢性髓细胞性白血病(CML)的患者的费城染色体编码的BCR-ABL融合疾病抗原。
“治疗”是指停止、延缓或阻止动物中疾病进展或预防动物中疾病发展。检测治疗是否成功的方法是已知的。例如,当疾病是实体瘤时,如果在施用有效量的包含使用本发明的各个实施方案中的方法产生的重组免疫球蛋白的治疗组合物之后肿瘤退化、转移降低、肿瘤大小减小和/或肿瘤细胞计数减少,则病情进展受到抑制、停止或延缓。
如本文所使用的,“有效量”是足以实现有益或所需结果(包括停止、减慢、停止、延缓或抑制动物中疾病进展或预防动物中疾病发展)的量或剂量。有效量将取决于,例如,待施用包含重组免疫球蛋白的治疗组合物的受试者的年龄和体重,症状的严重度和施用途径而不同,并且因此,施用基于个体而确定。通常,口服施用的成人每日剂量为约0.1至1000 mg,作为单一剂量或分份剂量而给予。对于连续的静脉内施用,该组合物可以0.01 ug/kg/min至1.0 ug/kg/min、期望0.025 ug/kg/min至0.1 ug/kg/min的范围内施用。
有效量可以一次或多次施用而进行施用。通过实例的方式,使用本发明的各个实施方案的方法产生的重组免疫球蛋白的有效量是足以改善、停止、稳定、逆转、减慢和/或延迟动物中疾病(例如,癌症)进展的量,或者是足以改善、停止、稳定、逆转、减慢和/或延迟患病细胞(例如,活检的肿瘤细胞)在体外生长的量。如在本领域中所理解,本发明的各个实施方案的重组抗体的有效量可能尤其根据动物病史以及其他因素诸如重组抗体的同种型(和/或剂量)而变化。
可凭经验决定施用组合物的有效剂量及进度,所述组合物包含本发明各个实施方案的非限制性重组抗体,且进行此类决定在本领域的技术范围内。本领域技术人员应了解:必须施用的剂量将根据(例如)将接受本发明各个实施方案的组合物的动物、施用途径、特定类型的所用组合物 (例如,组合物内重组抗体的同种型) 及其他向动物施用的药物而改变。当动物(例如,人患者)施用包含抗体的组合物时,选择适当剂量的抗体的指导见于抗体治疗性用途的文献中,例如Handbook of Monoclonal Antibodies, Ferrone等人编,Noges Publications, Park Ridge, N.J., 1985, ch. 22和pp. 303-357;;Smith等人,Antibodies in Human Diagnosis and Therapy, Haber等人编,Raven Press, New York, 1977, pp. 365-389。
根据上文提及的因素,单独使用的有效量的抗体的典型每日剂量的范围为每天约1μg/kg至至多100mg/kg体重或更多。通常,可使用以下剂量中的任一种:施用至少约50mg/kg体重;至少约10mg/kg体重;至少约3mg/kg体重;至少约1mg/kg体重;至少约750μg/kg体重;至少约500μg/kg体重;至少约250μg/kg体重;至少约100μg/kg体重;至少约50μg/kg体重;至少约10μg/kg体重;至少约1μg/kg体重或更多的剂量。在一些实施方案中,本文提供结合剂(例如,抗体)的剂量为约0.01 mg/kg至约50 mg/kg, 约0.05 mg/kg至约40 mg/kg, 约0.1 mg至约30 mg/kg, 约0.1 mg至约20 mg/kg, 约0.5 mg至约15 mg, 或约1 mg至10 mg。在一些实施方案中,剂量为约1 mg至5 mg。在一些可替代的实施方案中,剂量为约5 mg至10 mg。
通过在单一时间点或多个时间点单一直接注射至单一或多个部位,可实现本文所述的方法(包括治疗方法)。还可几乎同时施用至多个部位。施用频率可在治疗过程期间进行确定及调节,且施用频率基于实现所需结果。在一些情况下,本发明各个实施方案的重组免疫球蛋白的持续连续释放制剂可能是适当的。用于实现持续释放的各种制剂及设备是本领域中已知的。
包含本发明的重组抗体的组合物,可以配制用于任何合适方式的施用,包括例如,全身性、局部性、口、鼻、静脉内、颅内、腹膜内、皮下或肌肉内施用,或通过其他方法(诸如输注,其确保其以有效形式递送至血流)。还可通过隔离灌注(isolated perfusion)技术,诸如隔离组织灌注来施用抗体以发挥局部治疗作用。对于肠胃外施用,诸如皮下注射,载体优选包含水、盐水、醇、脂肪、蜡或缓冲剂。对于口服施用,可以采用任何上述载体或固体载体,诸如甘露糖醇、乳糖、淀粉、硬脂酸镁、糖精钠、滑石粉、纤维素、葡萄糖、蔗糖和碳酸镁。在一些实施方案中,对于口服施用,组合物的制剂对于消化道内的分解是耐受的,例如,如将本发明的各个实施方案的重组免疫球蛋白封装在脂质体中的微胶囊。可生物降解的微球(例如,聚乳酸聚乙醇酸酯)也可以用作用于本发明的治疗组合物的载体。合适的可生物降解的微球公开于,例如,美国专利号4,897,268和5,075,109中。
在本发明的一些实施方案中,组合物还可以包含缓冲剂(例如,中性缓冲盐水或磷酸盐缓冲盐水)、碳水化合物(例如葡萄糖、甘露糖、蔗糖或葡聚糖)、甘露糖醇、蛋白、多肽或氨基酸诸如甘氨酸、抗氧化剂、螯合剂诸如EDTA或谷胱甘肽、佐剂(例如,氢氧化铝)和/或防腐剂。或者,本发明的各个实施方案的非限制性组合物可以配制为冷冻干燥物。
在本发明的一些实施方案中,重组免疫球蛋白还可以被包裹到微囊中、胶体药物递送系统(例如,脂质体、白蛋白微球、微乳化剂、纳米颗粒和纳米囊)中或微乳剂中,其中微囊可例如通过凝聚技术或通过界面聚合法(例如,分别羟甲基纤维素或明胶微囊以及聚(甲基丙烯酸甲酯)微囊)来制备。这种技术描述于Remington's Pharmaceutical Sciences,第18版,A. Gennaro编辑,Mack Publishing Co., Easton, Pa., 1990;和Remington, The Science and Practice of Pharmacy 第20版. Mack Publishing, 2000。为了提高本发明各个实施方案的重组免疫球蛋白的血清半衰期,可以将拯救受体结合表位插入到抗体(尤其是抗体片段)上,如例如美国专利号5,739,277所述。如本文所使用的,术语“拯救抗体结合表位”是指负责提高IgG分子在体内的血清半衰期的IgG分子(例如,IgG1、IgG2、IgG3和IgG4)Fc区的表位。
在本发明的一些实施方案中,重组免疫球蛋白还可以配制成脂质体。包含重组免疫球蛋白的脂质体可利用本领域已知的方法制备,诸如Epstein等人, 1985, Proc. Natl. Acad. Sci. USA 82:3688; Hwang等人, 1980, Proc. Natl Acad. Sci. USA 77:4030; 以及美国专利号4,485,045 和4,544,545中所描述的方法。美国专利号5,013,556描述了循环时间延长的脂质体。特别有用的脂质体可利用包含磷脂酰胆碱、胆固醇和PEG-衍生的磷脂酰乙醇胺(PEG-PE)的脂质组合物的反相蒸发方法制备。通过一定孔径的滤膜以得到具有所需直径的脂质体可以挤出脂质体。此外,本发明各个实施方案的抗体(包括抗原结合结构域片段,诸如Fab′片段)可按照Martin等人,1982, J. Biol. Chem. 257:286-288描述的方法经由二硫键互变反应缀合至脂质体。本发明各个实施方案的重组抗体的施用包括局部(local)或全身性施用,包括注射、口服施用、颗粒枪或插入导管施用及局部(topical)施用。本领域技术人员熟悉表达载体的施用来获得外源蛋白在体内的表达。例如参见美国专利号6,436,908、6,413,942和6,376,471号。
在另一个方面,本发明提供降低动物中特征在于动物中存在疾病抗原的疾病发生可能性的方法,其中所述方法包括施用有效量的治疗组合物,所述治疗组合物包含本发明的各个实施方案的重组免疫球蛋白,其中由治疗组合物的免疫球蛋白特异性结合的抗原和疾病抗原是相同的。
疫苗制备通常描述于Vaccine Design ("The subunit and adjuvant approach" (eds Powell M. F. & Newman M. J., (1995) Plenum Press New York)。
在另一个方面,本发明提供了用于确定来自动物的抗体的氨基酸序列的试剂盒,其包括(a)用于从动物获得编码多种免疫球蛋白的免疫球蛋白链可变区的核酸序列的装置,和(b)用于将通过质谱分析的抗体的质谱信息与源自核酸序列的预测质谱信息相关联,以确定抗体的氨基酸序列的说明书。
本文公开的方法可以用来随着时间推移监测循环抗体,例如,在用抗原免疫的受试者中。在这些实施方案中,样品可以在多个时间点(例如,免疫之前和之后)从受试者采集,并且本文公开的方法用来鉴定每个时间点的循环抗体。可以在多个时间点比较循环抗体的组成,以确定接种的效力和/或时程。这可用于监测受试者受试者的免疫应答,和还有可用于疫苗开发中。
提供以下实施例以说明本发明,但非限制本发明的各个方面和实施方案。
实施例1
从特异性结合抗原的多克隆抗体群体鉴定个别抗体重链
在本实施例中,多种单克隆抗体源自特异性结合抗原的多克隆抗体群体。使用本发明的各个实施方案的方法,将从来自动物(其血清包含初始多克隆群体的)的核酸分子生成的遗传物质数据库的信息与来自单克隆抗体分析的肽数据库信息进行比较。
根据本文所述的方法使用对于兔免疫球蛋白链编码序列特异性的引物(参见,例如,在下面实施例6中的引物序列)从用抗原免疫的动物的脾细胞获得核酸序列。基于它们在数据库中的出现次数和每种CDR3在数据库中所有CDR3区中出现次数的百分比,对多克隆抗体的重链的CDR3区进行排序。表2显示前25个CDR3区和它们的频率。这些结果显示,多克隆混合物中的许多不同抗体中发现相同CDR3序列。该信息显示,特异性结合相同抗原的抗体经常在它们的CDR3区中(和推测在其他CDR区)共有序列。该信息显示,本文所述的方法将能够鉴定和分离将特异性结合抗原的那些免疫球蛋白链(或其片段)。
表 2
为了生成肽数据库,使用以下方法。
抗体的蛋白水解消化
将约10ug多克隆抗体群体浓缩且通过超滤(0.5ml 10K Amicon:Millipore)缓冲液交换。将初始体积首先浓缩,然后通过添加400ul 200mM Hepes(pH 8)进行交换。将样品通过在室温下在pH 8 Hepes中的80ul 8M尿素中重悬浮15 min而进行变性。将抗体在室温下在10mM DTT中还原40 min。用20mM IAA进行烷基化1小时。将尿素浓度降低至2M的终浓度。然后将样品五等分,且在37C分别用胰蛋白酶、Lys-C、Glu-C、胃蛋白酶或糜蛋白酶分别消化过夜。对于胃蛋白酶消化物,将样品浓缩,用3M醋酸交换,且在RT下消化1小时。将消化通过添加20%TFA进行猝灭,且使用Sep-Pack药液筒(Waters)进行纯化。将纯化的样品冻干且重悬浮,用于在LTQ Orbitrap Velos质谱仪上分析。
质谱
将通过用蛋白酶Lys-C、胰蛋白酶、糜蛋白酶、胃蛋白酶或Glu-C消化抗体级分产生的肽混合物(即通过单独用这些蛋白酶肽中的每一种消化抗体级分产生肽)通过个别使用LTQ Orbitrap Velos (Thermo-Fisher)杂合质谱仪通过LC-MS/MS进行分析。将样品使用FAMOS自动进样器(LC Packings)15 min上样到手浇熔融石英毛细管柱(125 um,内径18 cm)上,所述毛细管柱以MagicC18aQ树脂(5 m, 200 ?)填充且使用具有串联分流器(in-line flow splitter)的Agilent1100系列二元泵。色谱以400 nl/min的8–30%溶剂B使用二元梯度进行35 min(溶剂A,0.25%甲酸(FA);溶剂B,0.1%FA,97%乙腈)。随着肽从液相色谱柱洗脱进入质谱仪,将它们电离化,且将肽离子的质荷比测定,以生成MS1谱。质谱仪然后选择在该时刻洗脱且在过去35秒还没有进行MS2谱获取的20个丰度最高的肽离子,然后分离且片段化,进而,那些20个前体肽离子中的每一个产生20 个MS2产物离子谱。获取前体离子的一个MS1谱和随后以数据依赖方式获取20个MS2产物离子谱的完整周期在约1.6秒内完成,然后随着肽从液相色谱柱洗脱而连续重复。使用电荷状态筛选来排斥单电荷种类,且需要500个计数的阈值来触发MS/MS谱。在可能的情况下,LTQ和Orbitrap以平行处理模式操作。
数据库搜索和数据处理。
针对遗传数据库使用SEQUEST 算法来搜索MS/MS谱。搜索参数包括对于糜蛋白酶、Glu-C、Lys-C和胰蛋白酶的全酶特异性,和没有对于胃蛋白酶的酶特异性,其中亲本质量耐受性(parent mass tolerance)为50 p.p.m,对半胱氨酸的静态修饰为57.0214,对甲硫氨酸的动态修饰为15.9949。以±0.02 Da 的片段离子耐受性搜索HCD谱,而以±1 Da 的片段离子耐受性搜索CID谱。将肽经由目标诱饵法(target-decoy approach)过滤至1%肽FDR,其中使用线性判别函数基于参数诸如Xcorr、ΔCn和前体质量偏差对各肽评分。
结果
图4图示描述本实施例遵循的方法。将核酸序列使用Kabat规则(参见Kabat, E. A.等人, Sequences of Proteins of Immunological Interest, National Institutes of Health, Bethesda, Md., (1987)和Wu, T.T.和Kabat, E.A. J. Exp. Med. 132:211-250 (1970))进行分析以确定其中可变区和CDR3区(和其序列)位于序列内。接下来,通过质谱鉴定的多个单克隆抗体的重链的CDR3区的百分比覆盖率得到阐明。如下表3中所示,来自MS分析的多克隆抗体混合物的16种不同的肽序列得到鉴定,其中16个肽中的每个包括来自从动物收集的核酸序列的相应序列的全部(即,100%)CDR3区。
表 3
在表3中列出的肽中,还看到通过质谱观察到的最频繁出现的肽中的5个和源自来自核酸序列的信息的理论质谱一样。因此,该实验证明,通过将源自核酸序列的预测质谱(和基础序列)与从来自多克隆抗体的实际肽片段观察到的质谱进行比较和关联,很容易获得多个单克隆抗体序列(或其至少重链)。
实施例2
开发流感抗原特异性的重组人抗体
在2009-2010年的冬天,H1N1流感病毒株感染许多人,引起了死亡和永久性损伤。使用本发明的各个实施方案的非限制性方法,中和性抗体可以从以前暴露于类似病毒株的人克隆,且用作组合物来治疗目前患有该疾病的人患者。
因此,筛选已知在1918年的流感流行期间暴露于流感病毒的老年个体中可以中和1918年病毒的血清抗体的存在。为了做到这一点,遵循Yu等人, Nature 455:532-536, 2008(和在线补充;整体通过引用并入本文的论文和补充)中描述的方法。
鉴定其血清和/或血浆含有病毒中和性抗体的患者,且从这些患者采集血液,分离成细胞和血清和/或血浆。
根据标准方法(参见,例如,本文描述的方法)从血细胞分离B淋巴细胞,且从B淋巴细胞获得核酸分子。使用与人免疫球蛋白重链可变区(VH)和轻链可变区(VL)基因的上游和下游区域杂交的引物通过PCR扩增基因组DNA而这些细胞中分离编码免疫球蛋白链的核酸分子。制备这些引物的方法是免疫学领域中的标准方法(参见,例如,描述于Marks和Bradbury, "PCR Cloning of Human Immunoglobulin Genes" 于Antibody Engineering:Methods and Protocols, 248:117-134, 2003(通过引用并入本文)中的方法)。
通过使用这些PCR扩增引物获得的这些核酸分子用于填充遗传物质数据库。在遗传数据库内,将核酸序列使用标准软件包进一步操作,以确定由每个核酸序列编码的多肽的氨基酸序列,且编码的多肽用胰蛋白酶虚拟消化,其中从这种消化生成的预测得到的肽用于生成预测的质谱。
从患者的血液收集血清和/或血浆。血清和/或血浆中存在的抗体通过标准方法分离。例如,血清蛋白通过免疫球蛋白结合且非免疫球蛋白不结合的蛋白A琼脂糖柱。由于收集血液的个体不是新暴露于1918年流感病毒,所以通过将其血清抗体通过用1918年病毒(例如,减毒病毒或其片段)包被的第二柱而对其血清抗体进一步富集特异性结合1918年病毒抗原的抗体。接下来,用蛋白酶(例如木瓜蛋白酶)或化学蛋白切割试剂(其特异性切割免疫球蛋白铰链区附近,且去除非粘附的Fc部分)处理结合的抗体。最后,用胰蛋白酶处理结合的Fab或Fab2片段以生成肽片段,且然后所有片段使用液相色谱法分级,然后将片段通过质谱进行分析。使用算法诸如Sequest程序,观察到的肽的串联质谱与从患者的B淋巴细胞中提取的核酸序列的预测质谱相关联。使用该方法,可以鉴定在遗传物质数据库的独特免疫球蛋白链的预测氨基酸序列内发现的至少一种肽。然后将编码该免疫球蛋白链(或其可变区)的核酸序列从遗传数据库中检索出,且使用标准DNA合成方法进行合成。然后将合成的DNA序列亚克隆到表达载体中,然后将所述表达载体转染到CHO细胞中。接下来分离细胞产生的重组抗体,且测试与1918年病毒(或其片段)结合的能力。
然后将使用这种方法产生的重组抗体与药学可接受的载体组合,然后施用于受H1N1病毒感染的患者。因为这些重组抗体完全是人来源的,所以预期它们将不会被患者的免疫系统所排斥。
实施例3
获得核酸序列
该方案使用下一代测序(NGS),且基于454 NGS平台(FLX+、FLX或初级;由454 Life Sciences, Roche company, Branford, CT商售)。对于其他高通量NGS平台将需要稍作修改,且所述修改将基于NGS制造商的说明。
使用标准免疫方案(参见,例如,Coligan等人,同上)用目标抗原(一种或多种肽、重组蛋白、病毒、毒素等)免疫小鼠。通过针对特定抗原的血浆免疫球蛋白滴度监测免疫应答。可以根据标准方法收集血液、脾、骨髓、淋巴结或任何淋巴器官,且进行处理以分离B细胞。如果材料有限,这种分离程序也可以减少,且以使用针对动物的重链和轻链群体的免疫球蛋白可变结构域特异性PCR引物的直接RT-PCR程序替代。
当然,在一些实施方案中,核酸序列可以从生物材料直接测序(即,在测序前不经扩增)。用于从核酸序列直接测序的服务和试剂是商售的,例如,从Helicos BioSicences Corp. (Cambridge, MA)商售。例如,Helicos' True Single Molecule Sequencing可以直接测序DNA、cDNA和RNA。还参见美国专利号7,645,596; 7,037,687, 7,169,560;和出版物Harris等人, Science 320: 106-109, 2008; Bowers等人, Nat. Methods 6: 493-494, 2009;和Thompson和Milos, Genome Biology 12: 217, 2011(所有这些专利和出版物完整地通过引用并入本文)。
在一些实施方案中,在获得序列信息之前扩增核酸序列(例如,通过聚合酶链式反应)。
在一个非限制性实施例中,寡聚dT PCR引物用于RT-PCR中。在另一个非限制性实施例中,使用下述PCR引物进行基因特异性RT-PCR。在另一个实施例中,针对小鼠重链和轻链群体的PCR引物具有PCT公开号WO2010/097435(通过引用并入本文)中所述的序列。
在有或没有B细胞富集的情况下,然后根据标准程序(参见,例如,Ausubel等人,同上)将纯化的遗传物质(DNA或mRNA)进行RT-PCR。这是NGS测序运行之前遗传物质的文库制备阶段。逆转录(RT)反应可以应用寡聚dT或免疫球蛋白特异性引物以生成cDNA。聚合酶链式反应程序将应用免疫球蛋白特异性引物来从样品扩增(重排和/或表达的)重链和轻链的可变区。
下文进一步详细描述了这些方法。
文库制备
样品制备实施例:
在小鼠接受最后的抗原加强后分离血液、脾、骨髓、或淋巴结。如前面所述,通过Ficoll分离法分离单核细胞。然后Ficoll分离的细胞用PBS洗涤,计数,且速冻用于制备总RNA。
根据制造商的说明使用Qiagen RNeasy试剂盒(由Qiagen Inc., Hilden, Germany商售)从细胞分离总RNA,总RNA在-80℃下储存。
对于基因特异性RT-PCR或标准的RT-PCR(使用寡聚dT),可以使用下面的方案。
10uM CST小鼠RT-Ig引物或或寡聚dT 1ul
2.5ug总RNA (脾细胞) xul
10mM dNTP 2ul
无菌蒸馏水 至14ul。
将混合物在65℃下孵育5分钟,然后放置在冰上。
5x cDNA合成缓冲液 4ul
0.1M DTT 1ul
Invitrogen Thermoscript RT (15U/ul) 1ul。
轻轻混合内容物,且在65℃下孵育60 min
通过在85℃下加热5 min终止反应
cDNA准备用于制备文库。
然后将cDNA进行使用重链和轻链的CST 454特异性融合小鼠引物的PCR。引物具有以下序列:
小鼠454扩增子引物
重链(正向和反向引物)
κ链(正向和反向引物)
λ链(正向和反向引物)
在所有上述序列中,加下划线的序列用于454测序,粗体显示的序列是用于多重的条形码,常规字体序列是小鼠特异性序列。
引物用于如下扩增上述文库:
重链PCR:
CST454小鼠重链引物混合物 1ul
cDNA 1ul
2x Phusion母液混合物 12.5ul
H2O 10.5ul
轻链PCR:
CST454小鼠轻链引物混合物 1ul
cDNA 1ul
2x Phusion母液混合物 12.5ul
H2O 10.5ul。
PCR条件循环条件可以如下表4中:
表4
| 步骤 | 温度 | 时间(单位为分钟) |
| 1:变性步骤 | 98℃ | 01:30 |
| 2:变性步骤 | 98℃ | 00:10 |
| 3:退火步骤 | 60℃ | 00:30 |
| 4:延伸步骤 | 72℃ | 00:30 |
应用20个循环的步骤2-4。然后根据制造商的方案(参见,例如,Beckman Coulter Genomics' Agencourt AMPure XP系统的方案),PCR产物进行两次Agencourt Ampure DNA纯化(由Beckman Coulter Genomics, Danvers, MA商售)。
一旦制备PCR /基因文库,所有后续步骤将遵循emPCR和测序反应的454制备方案。参见如下出版物:454 Life Sciences Corp., a Roche Company, Branford, CT 06405出版,题为 "Sequencing Method Manual, GS Junior Titanium Series" (May 2010 (rev. June 2010))和"emPCR Amplification Method Manual – Lib-L, GS junior Titanium Series (May 2010 (rev. June 2010)),其两者都整体通过引用并入本文。
在这个阶段可以将多份样品组合成单一测序运行。它们将通过独特的条形码(或来自454平台的MID)进行区分。例如,条形码可以并入PCR引物中。
在一些实施方案中,遵循emPCR Amplification Method Manual – Lib-L, GS junior Titanium Series (May 2010 (rev. June 2010); 454 Life Sciences Corp.)。在一些实施方案中,接下来遵循Sequencing Method Manual, GS Junior Titanium Series" (May 2010 (rev. June 2010); 454 Life Sciences Corp.)。
测序数据可以FASTA文件(或任何标准的文件格式)产生,且储存在遗传物质数据库中。这些序列数据将用于生成预测的质谱数据库以分析从相同的动物血清和/或血浆免疫球蛋白生成的观察到的肽质谱。标准程序可用于做到这一点。在该实施例中,预测的质谱由Sequest软件包生成。
实施例4
从多克隆群体鉴定个别抗体链
本文描述的方法接下来用于从几种不同的多克隆群体鉴定个别抗体的序列。本实施例的方法示意性地显示在图2和图4中。
使用上述实施例2中描述的方法,将特异性结合三种不同抗原的三种不同的多克隆抗体群体纳入三个不同文库中。使用对于兔免疫球蛋白链编码序列特异性的引物进行使用上述454测序方法的深度测序,以获得三种不同的遗传物质数据库。
相应地,使用上述实施例3中描述的方法,遗传物质数据库用来生成三种不同的蛋白数据库。
第一抗原的结果显示于表5(轻链)和6(重链)中;第二抗原的结果显示于表7(轻链)和8(重链)中,且第三抗原的结果显示于表9(轻链)和10(重链)中。
表 5
表 6
表 7
表 8
表 9
表 10
表5-10显示通过质谱(CDR3肽)以高置信度(> 99%确定性)鉴定的肽,其对应于通过从动物的抗体所有组成成分的深度测序生成的序列(特别是CDR3区)。CDR3计数显示从匹配CDR3区的多克隆抗体混合物鉴定出肽的次数。CDR3覆盖率表明在质谱法鉴定的肽中出现的CDR3区(CDR3列中显示)的那些氨基酸相对于CDR3区的总氨基酸的百分比。总肽代表对应于通过深度测序确定的全长可变区序列的通过质谱法鉴定的序列的肽的总数。独特的肽代表对应于通过深度测序确定的全长可变区序列的通过质谱法鉴定的序列的独特肽的数目。
实施例5
在另一个实施例中,可以使用以下方案来生成核酸序列和多克隆抗体。结果显示使用这些方法成功生成了抗原特异性的抗体。
在这些方案中,用免疫原性P-ERK抗原免疫小鼠。可以使用以下方法生成遗传物质数据库和肽数据库。
I. 遗传物质数据库:
细胞分离。
来自免疫小鼠的脾脏使用注射器和21G针以5 mL RPMI/10%FCS冲洗5次。将细胞冷冻在90% FCS/10% DMSO中。从每个脾分离总共50-100 x 10^6个细胞。
RNA分离和cDNA合成。
根据制造商的方案,使用QIAshredder (Qiagen目录号79654)和RNeasy微小试剂盒(Qiagen, Hilden, Germany;目录号74104)从脾细胞中分离总RNA。根据标准的下一代测序方案将RNA在柱上DNAse处理。使用ND-1000分光光度计(NanoDrop;由Thermo Scientific, Wilmington, DE商售) 测定总RNA浓度。
分离的RNA用于使用Thermoscript RT-PCR系统(Invitrogen (Life Technologies的部分), Carlsbad, CA目录号11146-024)通过逆转录合成第一链cDNA。根据制造商的方案使用1.5ug RNA和寡聚dT引物合成cDNA。
VH和VL扩增。
使用两步PCR反应来扩增VH和VL基因。简并的有义和反义引物的混合物用于第一轮PCR,且一组通用引物用于第二轮PCR。由于使用大量有义简并引物,将重链PCR分成8个独立的反应。使用的引物序列如下显示。
第一轮引物,通用尾部加下划线
重链有义引物:
重链反义引物:
κ链有义引物:
κ链反义引物:
第二轮引物,通用尾部加下划线
重链或轻链有义引物:
重链反义引物:
轻链反义引物:
在上述序列中,下划线和斜体序列用于2步PCR扩增,加下划线的序列用于454测序,粗体显示的序列是454关键,小写序列是用于多重的条形码,且常规大写字体序列是小鼠特异性序列。
使用如表11中概述的上述引物设置PCR反应。
表11 (第一轮重链PCR设置)
对于第一轮,50 μL重链PCR反应含有0.2μΜ各有义引物(5条有义引物/每个反应)和0.2μΜ各反义引物(4条反义引物/每个反应)、10 μL 5x Phusion HF反应缓冲液(Finnzymes (Thermos Scientific的部分),目录号F-518)、1 μL cDNA、0.2 μM dNTP (NEB, 目录号N0447)、1 μL Phusion Hot Start II DNA聚合酶(Finnzymes, 目录号F-549L)和28 μL RT-PCR级水(Ambion (a Life Technologies company), Austin, TX, 目录号AM9935)。对于第一轮,50 μL轻链PCR反应含有0.2μΜ有义引物和0.2μΜ反义引物、10 μL 5x Phusion HF反应缓冲液(Finnzymes,目录号F-518)、1 μL cDNA、0.2 μM dNTP (NEB, 目录号N0447)、1 μL Phusion Hot Start II DNA聚合酶(Finnzymes, 目录号F-549L)和35 μL RT-PCR级水(Ambion, 目录号AM9935)。PCR热循环程序如下: 98℃持续2 min;15个循环(98℃持续0.5 min,55℃持续0.5 min,72℃持续1 min);72℃持续5 min;4℃储存。根据制造商的方案,使用DNA纯化和浓缩器-5试剂盒(Zymo Research Co., Irvine, CA, 目录号DR014)纯化PCR产物。
对于第二轮,50 μL重链PCR反应含有0.2μΜ通用有义引物和通用反义引物、10 μL 5x Phusion HF反应缓冲液(Finnzymes,目录号F-518)、10 μL纯化的第一轮PCR产物、0.2 μM dNTP (NEB, 目录号N0447)、1 μL Phusion Hot Start II DNA聚合酶(Finnzymes, 目录号F-549L)和19 μL RT-PCR级水(Ambion, 目录号AM9935)。PCR热循环程序为: 98℃持续2 min;10个循环(98℃持续0.5 min,55℃持续0.5 min,72℃持续1 min);72℃持续5 min;4℃储存。对于第二轮,50 μL轻链PCR反应含有0.2μΜ通用有义引物和通用反义引物、10 μL 5x Phusion HF反应缓冲液(Finnzymes,目录号F-518)、10 μL纯化的第一轮PCR产物、0.2 μM dNTP (NEB, 目录号N0447)、1 μL Phusion Hot Start II DNA聚合酶(Finnzymes, 目录号F-549L)和19 μL RT-PCR级水(Ambion, 目录号AM9935)。PCR热循环程序为: 98℃持续2 min;8个循环(98℃持续0.5 min,55℃持续0.5 min,72℃持续1 min);72℃持续5 min;4℃储存。根据制造商的方案,使用AMPure XP (Agencourt; Beckman Coulter Genomics, Brea, CA, 目录号A63881)纯化PCR产物,且使用Agilent 2100 BioAnalyzer进行分析。
然后将PCR产物的序列翻译成预测的氨基酸序列,然后将所述预测的氨基酸序列理论消化(例如,用蛋白酶和/或化学蛋白切割试剂),以产生虚拟的肽片段。然后这些虚拟的肽片段用于生成预测质谱。
II. 从多克隆抗体的肽片段生成实际质谱:
从动物的血清和/或血浆(例如,从获得核酸序列的动物的血清和/或血浆)纯化多克隆抗体。为了纯化抗体,使用以下方法:
蛋白-G纯化:
将1mL蛋白-G磁珠(Millipore (Billerica, MA), 目录号LSKMAGG10)添加到四个15 ml锥形管(Falcon (BD Biosciences, Franklin Lake, NJ),目录号352097)中的每一个中。每个管中的珠粒用10mL磷酸盐缓冲盐水pH7.4, 0.05% Tween-20 (PBST)洗涤两次,且用10mL PBS洗涤三次。将来自三只小鼠(ID 1262-2, 1262-4, 1263-4)的血清合并在一起,在PBS中稀释10倍至6ml终体积。然后将1.5ml合并且然后将稀释的血清添加至每个试管中的珠粒,且在4℃下孵育过夜。将流通物收集,且再投入通过纯化过程两次。收集流通物后,每管用10mL PBST洗涤两次,且用10mL PBS洗涤三次。然后每管在4℃下用0.5mL 0.1M pH 2.7甘氨酸孵育30分钟以洗脱IgG。洗脱重复5次。所有洗脱物用1M Tris pH 8.5中和,针对PBS透析过夜,且用ND-1000分光光度计(Nanodrop)测定蛋白浓度。总共纯化2.5mg IgG。
抗原柱制备:
5.0mL新鲜的链霉抗生物素蛋白(SA)磁珠(Pierce,目录号88817)用10mL PBS洗涤三次,且在4℃下与稀释在5.0 mL PBS中的105 uL 20mg/ml生物素p-ERK肽(由Cell Signaling Technology, Inc., Danvers, MA.商售的目录号1150的生物素化的形式)孵育过夜。弃去流通物,珠粒用10mL PBS洗涤三次,且等分至10个低结合1.7mL管(Axygen (Union City, CA), 目录号MCT-175-L-C)。将等分的珠粒置于磁性机架(Invitrogen, DynaMag)上,去除PBS,然后添加稀释的血清。
抗原特异性纯化:
将来自上述的蛋白-G纯化的IgG添加至与生物素P-Erk肽偶联的SA-磁珠。在4℃下过夜孵育后,收集流通物,且用含PBS的缓冲液洗涤珠粒。
IgG然后用5份的1.5mL 0.1M甘氨酸pH 3.5洗脱,然后用5份的1.5mL 0.1M甘氨酸pH 2.7洗脱,然后用5份的1.5mL 0.1M甘氨酸pH 1.8洗脱,且用1M Tris pH 8.5中和。洗脱物使用以p-ERK –BSA肽包被的96孔板测定P-ERK(即,磷酸化的ERK激酶,用于免疫小鼠的抗原)反应性。具有活性的级分通过ELISA(Thermo, 目录号23300)定量,且使用以20uM U0126处理1小时或200nM十四碳酰基 - 佛波醇-肉豆蔻酸(TPA)处理15分钟的Jurkat T细胞(例如,由美国典型培养物保藏中心或ATCC,Manassas, VA商售)的裂解物通过Western印迹测定 p-ERK反应性。具有最干净的p-ERK-反应性的级分通过质谱分析。
质谱分析
含抗体的级分用至少一种蛋白酶(例如,胰蛋白酶)和/或至少一种化学蛋白切割反应消化,且获得的肽使用质谱进行分析。用于分析肽的质谱分析方法是标准的,且以前已经详细描述过。(参见例如,美国专利号7,300,753; Geiger等人, Nature Methods 7: 383-385, 2010; Elias和Gygi, Nature Methods 4: 207-214, 2007; Keshishian等人, Molecular and Cellular Proteomics 6: 2212-2229, 2007, 其中所有均整体通过引用并入本文)。
如上所述(参见例如,实施例3),使用遗传物质数据库中的信息作为参考分析质谱。为了做到这一点,将MS2谱收集,然后使用标准计算程序(其为每个MS2谱(甚至当它不是优质谱或优质匹配时)寻找匹配)与从参考序列(即,从遗传物质数据库)预测的MS2谱逐个关联。这样的程序是商售的。例如,Sequest软件可以作为Sorcerer软件包的部分从Sage-N Research, Inc. (Milpitas, CA)获得。被鉴定为优质谱或与遗传物质数据库良好匹配的谱被映射到来自遗传物质数据库的参考序列。如果肽MS2可以被映射到遗传数据库的多于一个不同组分,那么还不清楚何种组分存在于结合抗原的多克隆抗体级分中,因为它可能是那些鉴定组分中的一种或多种。因此,重复该过程,随着重复,可以收集证据以显示,一些组分与优于其他的收集MS谱相关。换言之,其可变区序列中的一些被观察为通过抗原结合富集后的MS2谱。假定这些元件编码真正的抗原结合抗体,因此构建它们的序列(例如,在合成的寡核苷酸生成仪上),克隆进表达载体(例如,来自Invitrogen的pcDNA3.1),在细胞中表达,且检测抗原结合。
结果
如图5中显示,来自肽片段的实际质谱结果与来自核酸序列的理论质谱信息的关联允许鉴定重和轻链片段的序列。显示了就质谱覆盖率而言具有最高置信度的那些肽和与核酸序列的相关性。合成编码包含实际肽片段的全长链的核酸序列,且克隆到重组表达载体中。通过随机配对,将重链和轻链组合,在细胞中一起表达(即生成)重组抗体(参见,例如,美国专利号4,816,397;4,816,567;和美国专利申请号20110045534中的方法)。图6是显示使用pERK-包被板的ELISA实验的结果。如所见,图5中鉴定的一些链的配对导致产生能够特异性结合p-ERK-包被板的抗体(在图6中以黄色显示的阳性抗体,且阳性肽以红色显示在图5中)。
令人惊讶的是,这些结果显示,单独的肽存在频率和单独的CDR3计数频率都不能预测特异性结合抗原的具体抗体链的使用。例如,轻链核酸序列参考编号G623FKB01A3GC7与来自LC-MS/MS (液相层析、串联质谱)分析的235个肽相匹配,轻链核酸序列参考编号G623FKB01AXJ1C具有在单次NGS运行中出现1068次的序列(参见图5,下表)。然而,当与重链组合时,这些实际上都不能形成可以特异性结合pERK抗原的抗体。该结果是非常令人惊讶的,且显示,Reddy等人,Nature Biotechnology 28(9):965-969, 2010的方法(其仅依赖于来自NGS分析的核酸序列频率)已经错过了真正的抗原结合序列。因此,本文所述的方法可以用于可靠地鉴定和分离特异性地结合至选择的抗原的抗体。
实施例6
根据本文所述的方法生成抗原特异性的兔抗体。为了做到这一点,遵循以下方案。
兔脾细胞RNA纯化
p-MET抗原(Cell Signaling Technology, Inc., Danvers, MA 目录号1645)用于使用标准方法免疫兔。在最终的抗原注射(加强)后,处死具有抗原特异性血清(即血清含有特异性结合免疫抗原的多克隆抗体)的经免疫的兔。收集50 ml血液,且收集脾脏或其他淋巴器官。10百万个脾细胞用于纯化RNA。旁边还设置来自50 ml收集的血液的血清和/或血浆用于抗原特异性抗体亲和纯化。
遵循制造商的方案使用Qiagen's RNeasy试剂盒(Qiagen 目录号74104)从脾细胞纯化RNA。通过引入DNase I消化步骤进行在柱上DNase I处理以消除污染的基因组DNA。RW1缓冲液洗涤后,将RDD缓冲液中稀释的DNase I (Qiagen 目录号79254)应用于RNA纯化柱,且在室温下孵育20分钟。然后将柱再一次用RW1缓冲液洗涤,随后用RPE缓冲液洗涤两次,且用30或50μ1水洗脱RNA。通过在Nanodrop分光光度计(Thermo Scientific)上在波长450nm处测量的吸光度测定RNA的浓度。
cDNA合成和通过PCR生成 扩增子
首先如下所示使用Invitrogen的Thermoscript逆转录酶(Invitrogen 目录号12236-022)逆转录从兔脾细胞分离的RNA:
DNase处理的RNA: 5 uL
寡聚dT引物(50uM): 1 uL
dNTP's (10mM): 2 uL
dI H2O: 4 uL。
在65℃下孵育5分钟,然后放置在冰上2分钟,然后添加下列:
5X cDNA缓冲液: 4 uL
0.1mM DTT: 1 uL
RNAse OUT: 1 uL
dI H2O: 1 uL
ThermoScript: 1 uL。
将混合物在50℃下孵育1小时,随后在85℃下进行5分钟的热灭活步骤。最后,通过添加1μl RNase H (Invitrogen (Carlsbad, CA), 目录号18021-071)且在37℃下孵育20分钟而从cDNA去除互补的RNA链。
用于测序的重链、κ和λ链可变区的扩增子如下通过PCR生成。
重链融合引物:
反向
正向
κ链融合引物
反向
正向
λ链融合引物
反向
正向
在上述序列中,加下划线的序列用于454测序,粗体显示的序列是454关键,小写序列是用于多重的条形码,且常规字体序列是兔特异性序列。
PCR扩增使用Finnzyme's Phusion Hot Start II聚合酶(Thermo Scientific目录号F-540S)进行,其中反应混合物和条件设置如下:
反应混合物:
cDNA: 2.5 uL
5X缓冲液GC: 5 uL
10 mM dNTP混合物: 0.25 uL
Phusion HotStart II: 0.25 uL
引物(正向+反向) 30 uM: 0.25 uL
水: 16.75 uL
PCR程序:
步骤1 98℃ – 1.5分钟
步骤 2 98℃ – 10秒
步骤3 60℃ – 30秒
步骤4 72℃ –30秒
步骤5 重复步骤2至4,20次
步骤6 72℃ – 2分钟
步骤7 – 保持。
为了确保不存在来自任何试剂中的污染模板的任何虚假扩增,对于每种混合物设置一式两份的反应(对于重链4个独立的反应,对于每种轻链一个反应),其中用水取代cDNA模板。将这些无模板的阴性对照反应与含有模板的样品同时运行。完成PCR程序后,当将模板添加至反应但在cDNA存在的情况下,3μ1每个反应(包括阴性对照)在1.5%TAE琼脂糖凝胶上通过电泳分析扩增子的存在。图7显示这些凝胶电泳的结果。
扩增子纯化、分析、定量、且制备用于454测序
为了消除PCR样品中的过量引物和/或引物二聚体,遵循制造商的方案(000387v001)使用Agentcourt Ampure磁珠(Beckman Coulter 目录号A63881)纯化扩增子。然后通过遵循制造商的方案在Agilent 2100生物分析仪上使用高灵敏度DNA芯片(Agilent Technologies 目录号5067-4626)分析Ampure纯化后洗脱的扩增子的纯度和不存在任何污染的DNA种类。
一旦扩增子的纯度得到验证,则如制造商的方案中所述使用Quant-iT PicoGreen dsDNA测定试剂盒(Invitrogen 目录号P7589)在荧光计上定量DNA的浓度。试剂盒中提供的λ DNA用作浓度标准品,用所述浓度标准品从100ng/孔至1.56ng/孔生成标准曲线。一式两份测定在TE缓冲液中100倍稀释的每种扩增子的荧光,且根据标准曲线的线性部分测定DNA的浓度。所有荧光测量在黑色96孔板中进行。如果荧光值在标准曲线的线性范围之外,则以更大或更小的稀释度对样品进行重新测量,以捕获落在线性范围内的荧光值。使用每种链类型的碱基对的近似大小(重-540bp,κ-485bp和λ-510bp),下式用来确定浓度:
每种扩增子的浓度(分子数/μl) =
[样品浓度(ng/μl)*6.022x1023]/[656.6x109*扩增子长度(bp)]
将每种扩增子归一化到1 X 107个分子/μl,然后以3:3:1(体积)的重链:κ链:λ链的比例混合,涡旋,且最终1:10稀释,以获得1x106个分子/μl的最终浓度的混合物。
乳液PCR扩增、珠粒富集、珠粒计数和测序
遵循454公开的方案进行乳液PCR:"emPCR Amplification Method Manual – Lib-L" (Edition: May 2010 (Rev. April 2011, 其整体通过引用并入本文),其具有以下修改:
部分3.1.3 步骤2)
| 试剂 | 体积(μl) |
| 分子生物学级水 | 458 |
| 添加剂 | 515 |
| 扩增混合物 | 270 |
| 扩增引物 | 32 |
| 酶混合物 | 70 |
| PPiase | 2 |
| 总计 | 1347 |
一旦富集测序珠粒,根据"emPCR Amplification Method Manual – Lib-L"的步骤3.7,用以下设置在Beckman Coulter's Z2 Particle Counter上对珠粒计数:
孔径(Aperture): <100 μm>
孔径Kd(Aperture Kd): <60.04>
设置上截止值: <30.00μm>
设置下截止值: <10.00μm>
计数模式: <between>
定量体积: <0.5 ml>
分辨率: <256>
珠粒的浓度计算为:
珠粒的浓度 = [来自颗粒计数的平均读数 * 4] 珠粒/μl
遵循GS FLX+或GS Junior的454测序方案在454测序仪(Roche)上对从乳液PCR富集的珠粒进行测序。
如上对于小鼠所述生成从免疫的兔的血清收集的多克隆抗体的肽片段(参见,例如,实施例6)。简而言之,使用下面的方案。
肽亲和纯化兔IgG
1. 重悬浮肽亲和树脂,取0.4 ml浆料放入新柱(Bio-rad, 731-1550, 0.8x4cm),这应使0.2 ml纯化树脂沉降。如果必要,制备等体积的空白树脂或无关的肽亲和树脂的对照柱。空白树脂在缀合过程中没有用肽制备。
2. 用10ml PBS洗涤柱,且让它完全流尽。
3. 上样蛋白A纯化的总IgG。首先将柱加盖,且用石蜡包封。添加3-5ml总IgG。将顶部加盖,且用石蜡包封。
4. 在RT下在滚筒上旋转15 min。
5. 收集流通物。首先将顶部去盖,然后底部去盖,让柱完全流尽。
6. 用10ml PBS洗涤3次(洗涤柱壁,以确保所有树脂包装在底部)。
7. 用10ml 1x RIPA洗涤。
8. 用10ml PBS(pH7.4)中的20%乙腈洗涤。
9. 用10ml PBS(pH7.4)中的60%乙二醇洗涤。
10. 用10ml PBS(pH7.4)中的2.0M NaCl洗涤。
11. 用5ml 0.1M甘氨酸pH3.5洗脱,立即用70ul 1M Tris pH8.5中和。
12. 用5ml 0.1M甘氨酸pH2.7洗脱,立即用300ul 1M Tris pH8.5中和。
13. 用5ml 0.1M甘氨酸pH1.8洗脱,立即用800ul 1M Tris pH8.5中和。
14. 使用兔IgG ELISA板(由Molecular assay/ELISA group提供)测定所有或部分目标级分的IgG浓度。
15. 可以使用ELISA和/或Western印迹评价抗原特异性活性。也可以将所有级分均一化至相同浓度后评价比活性。
16. 纯化的抗体材料准备处理用于LC-MS/MS
如上所述对来自纯化的抗体(即,消化纯化的抗体且分析肽)进行液相色谱 - 串联质谱(LC-MS/MS)分析。将获得的质谱与基于遗传物质数据库中的信息的理论质谱数据相关联。
如图8中所示的表格显示,通过将肽的实际(即,观察的)质谱与来自核酸序列的理论质谱信息相关联而鉴定多种重和轻链肽。这些肽的出现频率显示在表中最右侧道中。基于其CDR3的覆盖率(在大多数情况下,100%)选择这些链,从遗传物质数据库检索潜在的核苷酸序列且进行合成。将六条重链与五条轻链随机组合(图8中以红色显示),且获得的抗体使用ELISA(与抗原包被板)和Western印迹分析(针对未经处理的(-泳道)或人生长因子处理的(+泳道)Hela细胞)进行检测,其中HGF处理的细胞已知表达p-MET抗原。Western印迹分析的结果显示于图9。p-MET特异性抗体(由Cell Signaling Technology, Inc., Danvers, MA商售,目录号3126)用作对照。根据本文所述方法生成且显示与细胞裂解物中的抗原高特异性结合的抗体在图8中以粗体红色显示(即,重链参考编号GXRYQP201BIQD2和GXRYQP201A97DZ和轻链参考编号GXRYQP201A291T和GXRYQP201BRIWK和GXRYQP201ALDF5)。注意,图9仅显示本实施例中生成的与抗原特异性结合6种不同抗体中的两种(即,图9仅显示使用与GXRYQP201A291T轻链和GXRYQP201BRIWK 轻链偶联的GXRYQP201BIQD2重链的两种抗体。
同样,如用小鼠抗体观察的,具有最高频率的链没有导致形成抗原 - 特异性抗体(比较重链GXRYQP201A1C3B(其具有9.12%的频率,但不特异性结合抗原)和重链GXRYQP201 BIQD2(其具有仅0.19%的频率,但的确特异性结合抗原)。
实施例7
本实施例描述了使用本文上述方法从四种不同抗原免疫的兔和一种额外的不同抗原免疫的小鼠生成单克隆抗体(表12),进一步表明本方法在至少两种实验动物物种中的稳健性和重现性。
表12. 通过ELISA和Western印迹(WB)测试的通过NGS/LC-MS/MS平台鉴定的针对多种目标的功能上相关的单克隆抗体。
用与钥孔血蓝蛋白(KLH)缀合的人孕酮受体A/B特异性(PR A/B)肽免疫新西兰白兔。筛选每只动物的粗血清中的抗原特异性抗体活性,以选择对PR A/B具有最高ELISA和Western印迹信号的兔。从20 mL血液收获来自该动物的血清,且从脾B细胞获得RNA。使用蛋白A琼脂糖柱从血清中分离总γ免疫球蛋白(IgG),且使用由与琼脂糖珠缀合的抗原特异性肽组成的定制柱通过亲和色谱纯化抗原特异性多克隆抗体。结合的IgG用PBS充分洗涤,然后进行用逐步酸性缓冲液(pH 3.5、pH 2.7和pH 1.8)的顺序洗脱(图10a)。收集来自每次洗脱的级分,中和,且通过PR A/B表达细胞系T47D和PR A/B阴性细胞系HT1080的裂解物的抗原特异性ELISA和Western印迹进行筛选(图10a)。发现当多克隆级分浓度匹配时,PR A/B Western印迹特异性活性在pH 1.8级分中高度富集,在较小程度上在pH 2.7级分中富集,且在pH 3.5级分中检测不到。因此pH1.8级分用于LC-MS/MS分析。
为了通过NGS生成Ig V区序列的定制数据库,从显示对PR A/B 的强特异性活性的相同动物收获的总脾细胞分离RNA。使用用来扩增整个V区的兔Ig特异性γ和κ链引物来生成Ig重链和轻链可变区扩增子。引物含有条形码,且遵循为Roche 454 NGS平台的454钛融合引物设计的具体要求。为了增加收集的V区序列的数量,我们组合了由γ链和κ链组成的三次454 GS Junior测序运行,其导致产生总共80,000个通过的过滤读取,其中44,363个含有整个V区且提供了下述蛋白组学方法的基础。收集的序列包括具有遵循高斯分布的不同长度的5,279个独特的γ链互补性决定区3(CDR3)序列和11,681个独特的κ链CDR3序列。与以前数据相一致,该兔优先使用以重链VDJ重排的VH1 (V1S69+ V1S40 >64%)和随后的VH4 (V1S44+ V1S45 ~30%) (Becker 等人, Eur J Immunol 20: 397-402, 1990, Knight, Annu Rev Immunol 10: 593-616, 1992, Mage等人, Dev Comp Immunol 30: 137-153, 2006)。
接下来,pH 1.8级分基于其以前的活性通过LC-MS/MS进行检查(图10a)。为了使序列覆盖率最大化,均分5ug多克隆抗体,且分别通过糜蛋白酶、弹性蛋白酶、胃蛋白酶和胰蛋白酶消化。使用Orbitrap Velos (Thermo Fisher)收获使用45 分钟梯度的总共四次LC-MS/MS运行,产生平均10,000谱/每次运行(图10b)。为了评价假发现率(FDR),通过生成正向和反向序列的复合数据库(Elias等人, Nat Methods 4:207-214, 2007)来使用目标/诱饵方法,且使用SEQUEST (Yates等人, Anal Chem 67:1426-1436, 1995)程序搜索每次LC-MS/MS运行。使用当可能时考虑酶特异性(糜蛋白酶/胰蛋白酶)的线性判别分析(Huttlin等人, Cell 143:1174-1189, 2010)将肽谱匹配(PSM)过滤到≤ 2%的最终FDR。使用该方法鉴定的高置信度重链CDR3肽的实例显示于图10c中。组合各次运行,且鉴定了总共2,356个V-区PSM,FDR为1.8%。
抗体V区序列的数据库与蛋白同种型的数据库类似。作为结果,使用通过LC-MS/MS的鸟枪法测序的常规方法(其中只有少数肽经常用来可靠地鉴定蛋白)对于在多克隆抗体混合物中鉴定抗体V区序列是不足的。此外,由于抗体V区序列可以变化少至一个氨基酸,高质量精度帮助提供PSMs中的额外置信度。将通过SEQUEST确定的具有质量误差≥-5和≤5ppm的每个V区PSM映射回整个V区数据库以解决PSM冗余和整个数据集的覆盖率(图10d)。重新映射后,对于每个V区序列确定总肽数、独特肽数量、谱共享(映射到序列的总肽/总V区PSM)、总V-区序列覆盖率和CDR3覆盖率。为了鉴定可能从多克隆混合物富集的具有高置信度的V区序列,在蛋白质组学分析中应用经验上严格的标准,包括:a) 高整体覆盖率(≥65%),b) 由于V区序列高度同源性的至少12个独特肽,和c)高超变区覆盖率,特别地CDR3的≥95%覆盖率。尽管V区序列可以单独使用一种蛋白酶进行鉴定,但是发现,因为V区序列中高度可变性连同多克隆混合物的不可预知的复杂性,使用多种蛋白酶来增加V区覆盖率是有利的。例如,如图10e中所示,来自不同蛋白酶的多个重叠的肽片段有利于鉴定重链和轻链序列两者的整个CDR3。鉴定可以映射至相同V区序列的来自多种蛋白酶的多次运行间独特的PSMs增加了谱计数,并且整个V区序列间的覆盖率提供了特定V区序列存在于多克隆混合物中的高置信度,并且进一步增加了NGS序列质量中的置信度(Kircher等人, Bioessays 32:524-536, 2010)。使用上述过滤标准,从pH 1.8洗脱级分总共鉴定了具有高置信度的10个γ链序列和8个κ链序列(表13)。
表13. 高置信度重链和轻链的鉴定。具有100% CDR3谱覆盖率和总体≥65%可变区覆盖率的重链和轻链得到鉴定,并且以通过总肽计数测量的置信度的顺序进行排序。还表示CDR3序列同一性和兔种系测定。选择重链和轻链用于基因表达、克隆和用于表征的组合抗体的表达。NGS排序表示在NGS数据库中对于每条链鉴定的给定CDR3序列的排序。*表示不可能鉴定到D基因。
尽管提供了亲和纯化的血清中存在的高置信度V区序列存在的证据,但是LC-MS/MS数据缺乏关于同源重链和轻链配对的直接信息,这是由于样品制备过程中的蛋白水解和二硫键的还原导致的。作为结果,除了通过NGS频率观察到的高排序重和轻序列外,表达重链和轻链配对的所有可能组合(对于总共80种抗体的8×10矩阵,在一次96孔板转染中),且通过ELISA筛选对PR A/B肽的抗原特异性结合活性。通过抗原特异性ELISA,总共12对重链和轻链对是阳性的(图11a)。然后通过Western印迹测试每种抗原特异性ELISA阳性克隆针对细胞裂解物中内源表达的PR A/B的特异性(图11b)。发现六个克隆特异性结合PR A/B (图11b);当在相同的抗体浓度测定时,两个克隆显示与初始多克隆混合物相比强得多的信号。在额外测定中进一步表征通过Western印迹阳性的抗原特异性克隆。一种单克隆抗体,克隆F9和克隆C1,在Western印迹和免疫组织化学(IHC)中表现出优异的信号和特异性(图11b-c),其还在流式细胞术(FC)和免疫荧光(IF)中特异性反应,其中多克隆混合物无法特异性反应(图11d-e)。与之相比,凭借其最高NGS排序选择的γ和κ链没有产生抗原特异性抗体。从最高NGS排序γ和κ链中没有观察到含CDR3的肽,且通过我们的蛋白组学方法没有以具有高置信度鉴定出来自30条最高排序γ或κ链的CDR3序列。不能排除,活性的缺乏可能是由于缺乏同源配对导致的,但是通过LC-MS/MS没有观察到这些链中的任一条这一事实表明最高排序的NGS链中没有一条针对抗原是特异性的。因此,在这些实验中,抗原特异性抗体不能仅仅依靠NGS排序来鉴定。
为了显现克隆多样性,在表13中显示的高置信度重和轻链V区序列上进行系统进化分析(Dereeper等人, Nucleic Acids Res 36:W465-469, 2008)。重或轻链的密切相关序列成簇为离散组。有趣的是,本报道中发现的所有PR A/B特异性单克隆抗体在系统进化树中紧密地成簇在一起,最可能是由于在免疫过程中从密切相关的B细胞的克隆扩增导致的。种系使用也支持该观察(表13)。在独立的实验中使用不同抗原进行了类似观察(Lin28A,图12)。
在本实施例中描述的实验中使用的方法如下。
动物的免疫和处理。使用钥孔血蓝蛋白缀合的源自每种人蛋白抗原不同区域的氨基酸序列的肽的混合物以四次单独剂量(每次隔开3周)通过皮内注射免疫新西兰白兔。将肽缀合至Imject马来酰亚胺活化的KLH(Thermo-Pierce)。以相同方式进行小鼠免疫,除了免疫途径为腹膜内且注射间隔2周。最终加强后3天采血。确认所需多克隆活性后,在安乐死时从每只动物收获整个脾脏。
兔和小鼠B细胞所有组成成分的下一代DNA测序。收获来自超免疫兔和小鼠的脾细胞,且裂解,用于遵循制造商的方案使用Qiagen's RNeasy试剂盒纯化总RNA。使用提供的方案用DNase I (Qiagen 目录号79254)在柱上处理RNA以消除基因组DNA。为了从这些材料生成待用454 Life Sciences平台(Roche)测序的重和轻链扩增子文库,如下进行RT-PCR。使用Thermoscript逆转录酶(Invitrogen 目录号12236014)以寡聚dT为引物从作为模板的脾细胞总RNA生成cDNA。对于兔IgG测序,用以下步骤使用Phusion? Hot Start II高保真DNA聚合酶(Finnzymes Oy, Finland)用序列特异性454融合引物(与5'末端上的前导序列杂交,且在3'末端上含有454测序平台的Lib-L形式中鉴定和条形编码所需的序列)扩增γ、κ1、κ2、和λ链的可变区:变性-98℃持续90秒;20个循环的[变性-98℃持续10秒,退火-60℃持续30秒;延伸-72℃持续30秒]。对于小鼠IgG测序,通过两步PCR方法生成重和轻链扩增子。在第一步骤中,使用作为有义引物的基因家族特异性简并寡核苷酸和与恒定区的开始处高度保守区杂交的反义引物(每条有义和反义引物在其5'端含有独特的适配子序列)的混合物扩增γ或κ链可变区(以与上述对于兔的相同条件进行15个循环)。来自第一轮的每个反应使用商售试剂盒(Qiagen目录号28104)进行柱纯化,然后在第二步骤中使用适配子序列特异性引物(其在3'末端上含有454测序平台的Lib-L形式中鉴定和条形编码所需的序列)通过额外10个循环(γ链)和8个循环(κ链)进一步扩增。对于任一种类,将每只动物的所有轻链扩增反应合并。遵循提供的方案使用Agencourt AMPure XP DNA纯化系统去除重链和轻链样品的过量引物。在Agilent Bioanalyzer 2100 (Agilent Technologies)上验证引物去除后扩增子合并物的质量和纯度,且使用Quant-iT PicoGreen dsDNA测定试剂盒(Invitrogen)在荧光计上准确定量DNA的浓度。Lib-L LV后,进行来自454 Life Sciences 的GS FLX Titanium Series系列方案,乳液PCR和珠粒富集。在Beckman Coulter Z2颗粒计数器上计数珠粒数量,且在454 GS Junior (Roche)上对文库测序。
抗原特异性IgG的亲和纯化。使用蛋白A琼脂糖珠粒(GE Healthcare)纯化来自超免疫兔(新西兰白兔)的血清的总IgG,然后在具有与琼脂糖珠粒共价偶联的免疫原肽的柱中旋转孵育15分钟。通过重力流,排尽未结合的级分,且用1x磷酸盐缓冲盐水(PBS)充分洗柱,以消除非特异性IgG。抗原特异性多克隆IgG合并物相继用pH 3.5和随后的pH 2.7和最后pH 1.8的0.1M甘氨酸/HCl缓冲液进行洗脱。每次洗脱立即用1M Tris缓冲液(pH 8.5)中和。来自超免疫小鼠血清的总IgG使用蛋白G磁珠(Millipore, 目录号LSKMAGG10)纯化,然后与固定在磁珠(Pierce, 目录号88817)上的免疫原肽在4℃下旋转孵育过夜。使用磁性管架(Invitrogen, 目录号12321D),用PBS充分洗涤珠粒,然后如对于兔IgG纯化所述用逐步酸性pH相继洗脱结合在柱上的抗体。
亲和纯化抗体的蛋白酶消化。多克隆抗体在20mM HEPES pH 8中的8M尿素中变性,然后在55℃下在10mM DTT中还原1小时。将还原的多克隆抗体冷却至室温(RT),且在20mM碘乙酰胺存在的情况下进行烷基化1小时。在37℃下以1:50的酶和底物比在20 mM HEPES pH 8.0中2 M尿素存在的情况下进行糜蛋白酶、弹性蛋白酶和胰蛋白酶消化过夜。在RT下以1:50的酶和底物比在3 M乙酸存在的情况下进行胃蛋白酶消化过夜。消化的肽如以前公开(Rappsilber等人, Anal Chem 75:663-670, 2003)通过STAGE-TIPS进行脱盐,且通过LC-MS/MS分析。
质谱。使用LTQ Orbitrap Velos (Thermo-Fisher)质谱仪进行LC-MS/MS。将样品使用Famos自动进样器(LC Packings)7 min上样到手浇熔融石英毛细管柱(125 μm,内径X 20 cm)上,所述毛细管柱以Magic C18aQ树脂(5 μm, 200 ?)填充且使用具有串联分流器(in-line flow splitter)的Agilent1100系列二元泵。色谱以400 nl/min的5–30%溶剂B使用二元梯度进行45 min(溶剂A,0.25%甲酸(FA);溶剂B,0.1%FA,97%乙腈)。在Orbitrap (300–1,500 m/z,以6x104的分辨率设置)中从以前的主谱以数据依赖的方式获得二十个MS/MS谱,自动增益控制(AGC)目标为106。使用电荷状态筛选来排斥单电荷种类,且需要500个计数的阈值来触发MS/MS谱。在可能的情况下,LTQ和Orbitrap以平行处理模式操作。
数据库搜索和数据处理。MS/MS谱使用SEQUEST算法(版本28 rev 12) (Yates等人, Anal Chem 67:1426-1436, 1995)针对定制杂合数据库进行搜索,所述数据库由21,932个全长γ V区序列和22,431个全长κ V区序列和γ和κ恒定区序列串联6,358种酵母蛋白(酿酒酵母,NCBI)和42种常见污染物(包括几种人角蛋白、胰蛋白酶和糜蛋白酶)所构成。由于V区序列是高度相关的,酵母蛋白质组将更多样的序列人工提供给参考数据库(Beausoleil等人, Nat Biotechnol 24:1285-1292, 2006),并且在过滤最终数据集之后提供了另一个置信度来源,因为过滤的数据不应该包括从酵母鉴定的肽。搜索参数包括对于糜蛋白酶和胰蛋白酶的部分特异性,和没有对于弹性蛋白酶和胃蛋白酶的特异性,其中质量耐受性(mass tolerance)为±50 ppm,对半胱氨酸的静态修饰为57.0214,对甲硫氨酸的动态修饰为15.9949。数据集中的错误发现率使用目标/诱饵方法(Elias等人, Nat Methods 4:207-214, 2007)来评估。使用线性判别分析(Huttlin等人, Cell 143:1174-1189, 2010)将数据集过滤到≤ 2%的FDR。尽管Orbitrap的质量精确度大大超过50ppm,但是当用更宽的前体离子耐受性搜索时,正确的肽鉴定导致小前体质量误差(± 1 ppm),而不正确的肽鉴定在整个50ppm窗口分布。因此,严格的前体质量滤器从数据集选择性去除许多不正确的PSM。
如文中所述进行获得后分析。简而言之,将源自V区序列的通过的肽重新映射到NGS Ig数据库。对于从糜蛋白酶和胰蛋白酶消化产生的肽,匹配限于预期切割而产生的那些(对于胰蛋白酶为KR,对于糜蛋白酶为YWFLMA)。CDR覆盖率通过使用由Kabat (Wu等人, J Exp Med 132:211-250, 1970)定义的规则鉴定CDR而确定。在所有情况下,覆盖率被定义为从高置信度肽鉴定的氨基酸总数除以成熟V区序列中氨基酸的数量。
鉴定的免疫球蛋白链的克隆、表达和表征。如下将通过亲和纯化的多克隆IgG合并物的质谱分析鉴定的γ和κ链进行克隆和表达。对于每种鉴定的链,合成编码FWR1至FWR4的整个可变结构域的核酸序列(Integrated DNA Technologies, Coralville Iowa)。使用重叠PCR,以使用核糖体跳跃机制来从单一开放阅读框生成两种多肽的病毒2A序列表达每种重-轻链组合排列(Doronina等人, Mol Cell Biol 28: 4227-4239, 2008, Donnelly等人, J Gen Virol 82: 1027-1041, 2001)。将从5'到3'顺序的轻链可变区和恒定区、来自Thosea asigna病毒的2A肽序列和重链可变结构域的单一开放阅读框盒克隆到分别含有符合读框的兔γ链前导序列和兔γ链恒定区、克隆位点的5'和3'端的CMV启动子驱动的哺乳动物表达质粒。使用聚乙烯亚胺用以这种方式装配的编码各轻-重链组合的质粒制备物转染HEK293(Boussif等人, Proc Natl Acad Sci USA 92: 7297-7301, 1995)。转染后2至5天通过使用免疫原肽作为包被抗原的ELISA筛选上清液中抗原特异性抗体的分泌,显示反应性的轻-重链排列进行进一步表征。对于小鼠抗体的表达,恒定区是小鼠IgG2a的。
通过ELISA、Western印迹、流式细胞术、免疫荧光和免疫组织化学表征多克隆抗体和单克隆抗体。ELISA、Western印迹、流式细胞术、免疫荧光和免疫组织化学的详细方案可以在Cell Signaling Technology Inc的网站上在线找到。Costar目录号3369认证的高结合聚苯乙烯96孔板用于ELISA。用于针对每种目标的ELISA分析的抗原是与用于免疫相同的肽。对于孕酮受体的抗体,对T47D (PR+)、MDA-MB-231细胞(PR-)和HT-1080 (PR-)细胞裂解物进行Western印迹,对T47D (PR+)和MDA-MB-231细胞(PR-)进行流式细胞术分析,对MCF-7细胞(PR+)(与MDA-MB-231细胞(PR-)相比)进行共聚焦免疫荧光分析,且对石蜡包埋的原发性人乳腺癌载片、T47D和石蜡包埋的MCF-7细胞(PR+)(与MDA-MB-231细胞(PR-)相比)进行免疫组织化学分析。对于磷酸化-p44/42 MAPK小鼠抗体,对用U1026 (Cell Signaling Technology, Inc. 目录号9903)或12-O-十四烷酰佛波醇-13-乙酸酯(TPA) (Cell Signaling Technology, Inc. 目录号4174)处理的Jurkat细胞的裂解物进行Western印迹。对于Lin28A抗体,对NCCIT、NTERTA、MES和IGROV1细胞系的总裂解物进行Western印迹,对NTERA (Lin28A+)和HeLa (Lin28A-)细胞进行共聚焦免疫荧光和流式细胞术分析。对于磷酸化-Met (pMet)抗体,使用用SU11274 Met 激酶抑制剂处理(pMet+)和未处理(pMet-)的MKN45细胞的裂解物。对于Sox1抗体,使用小鼠脑提取物(Sox1+)和NIH-3T3 (Sox1-)细胞的裂解物。
实施例8
在本实施例中,根据本文所述的方法生成对于乙型肝炎病毒表面抗原(HBsAg)特异性的人单克隆抗体。为了做到这一点,遵循以下方案以生成遗传物质数据库。多克隆抗体如下所述进行纯化,且如上对于小鼠和兔所述遵循质谱分析进行分析。
I. 生成核酸序列
抗原特异性、记忆性和总B细胞分离和RNA纯化
使用Ficoll梯度从收集在肝素真空管中的新鲜、完整的人血液分离外周血单核细胞(PBMC)。在底部含有20ml Histopaque 1077 (Sigma Aldrich目录号10771)的Greiner Leucocep 50ml锥形管(Sigma Aldrich 目录号Z642843)中,在顶部应用多达25ml血液,然后在室温下将试管以1500xg离心20分钟。白细胞(血沉棕黄层)使用无菌移液管收集,通过在50ml RPMI中重悬浮细胞而在RPMI培养基中洗涤两次,然后在4℃下以300xg离心10分钟。洗涤后,PBMC冻存在胎牛血清中的20% DMSO中或立即处理用于分离B细胞。
为了分离B细胞分离,遵循制造商的方案使用Invitrogen's Dynabeads Untouched B-细胞分离试剂盒(Invitrogen 目录号113-51D)使用阴性选择方法来从PBMC消除所有非B细胞。将获得的未标记的B细胞群体进一步处理以分离抗原特异性或记忆性B细胞。
为了分离抗原特异性B细胞,在室温下在旋转器上将总的未标记的B细胞与固定在链霉抗生物素蛋白磁珠(Pierce-Thermo Scientific目录号88816)上的生物素化的抗原孵育20分钟。然后含有任何抗原结合的B细胞的珠粒用1xPBS洗涤两次。洗涤的珠粒然后重悬浮在Qiagen's RNeasy试剂盒RLT裂解缓冲液(补充有1%β-巯基乙醇)中,用于分离RNA。
为了分离记忆性B细胞,使用用于CD27+和表面IgG+细胞分离的Miltenyi's MACS试剂盒(Miltenyi Biotec (Auburn, CA) 目录号130-051-601和130-047-501)从总的未标记的B细胞分离CD27+和表面IgG+细胞。为了同时分离CD27+和sIgG+ B-细胞,在孵育步骤过程中同时添加针对这两种细胞表面标志物的磁珠缀合的抗体。纯化后,将记忆性B细胞以300xg离心10分钟,然后如上对于RNA分离所述在用于RNA的RLT缓冲液中裂解。
遵循制造商的方案使用Qiagen's RNeasy试剂盒(Qiagen 目录号74104)从选择的细胞纯化RNA。通过引入DNase I消化步骤进行在柱上DNase I处理以消除污染的基因组DNA。RW1缓冲液洗涤后,将RDD缓冲液中稀释的DNase I (Qiagen 目录号79254)应用于RNA纯化柱,且在室温下孵育20分钟。然后将柱再一次用RW1缓冲液洗涤,随后用RPE缓冲液洗涤两次,且用30或50μ1水洗脱RNA。通过在Nanodrop分光光度计(Thermo Scientific)上在波长450nm处测量的吸光度测定RNA的浓度。
cDNA合成和通过PCR生成扩增子
首先如下所示使用Invitrogen的Thermoscript逆转录酶(Invitrogen 目录号12236-022)逆转录从记忆性或抗原特异性B细胞分离的RNA:
DNase处理的RNA: 5 uL
寡聚dT引物(50uM): 1 uL
dNTP's (10mM): 2 uL
dI H2O: 4 uL。
在65℃下孵育5分钟,然后放置在冰上2分钟,然后添加下列:
5X cDNA缓冲液: 4 uL
0.1mM DTT: 1 uL
RNAse OUT: 1 uL
dI H2O: 1 uL
ThermoScript: 1 uL。
将混合物在50℃下孵育1小时,随后在85℃下进行5分钟的热灭活步骤。最后,通过添加1μl RNase H (Invitrogen, 目录号18021-071)且在37℃下孵育20分钟而从cDNA去除互补的RNA链。
用于测序的重链、κ和λ链可变区的扩增子如下通过PCR生成。为了扩增重链,对于各cDNA样品使用下列引物运行4个独立的反应(每一个对于VH1和7;VH2、5和6;VH3;和VH4的基因家族是特异性的),以保持在B细胞合并物中VH基因转录物频率的自然分布。对于κ和λ链扩增,对于各cDNA样品运行每条链的每一反应。对于各反应,使用正向引物的等摩尔混合物与如下所示相同浓度的反向引物。使用对于Lib-L 平台的454测序(Roche)相容的融合引物进行扩增。反向引物设计为与每条链的恒定区的5'末端杂交。这些引物含有Lib-L引物B和MID序列,从而使得测序读取从各恒定区的最5'末端(以相反的意义)开始,且进入可变区的3'末端。对于重链和κ链,对于各MID使用单一反向引物,而对于λ链,对于各MID需要两条不同的反向引物。
PCR扩增使用Finnzyme's Phusion Hot Start II聚合酶(Thermo Scientific目录号F-540S)进行,其中反应混合物和条件设置如下:
反应混合物:
cDNA: 2.5 uL
5X缓冲液GC: 5 uL
10 mM dNTP mix: 0.25 uL
Phusion HotStart II: 0.25 uL
引物(正向+反向) 30 uM: 0.25 uL
水: 16.75 uL。
PCR程序:
步骤1 98℃ – 2分钟
步骤2 98℃ – 10秒
步骤3 60℃ – 30秒
步骤4 72℃ –30秒
步骤5 重复步骤2至4
步骤6 72℃ – 2分钟
步骤7 – 保持。
当分别扩增从记忆性B细胞或从抗原特异性B细胞生成的cDNA模板时,对于重链扩增,运行25或30个循环(步骤5重复24次或29次),对于κ和λ链,运行20或30个循环,因为对于每条链从抗原特异性B细胞的cDNA的充分扩增需要5个额外循环。为了确保不存在从任何试剂中的污染模板的任何虚假扩增,对于每种混合物设置一式两份的反应(对于重链4个独立的反应,对于每种轻链一个反应),其中用水取代cDNA模板。将这些无模板的阴性对照反应与含有模板的样品同时运行。完成PCR程序后,当将模板添加至反应但在cDNA存在的情况下,3μ1每个反应(包括阴性对照)在1.5%TAE琼脂糖凝胶上通过电泳分析扩增子(对于重链约540bp,对于κ链约485bp且对于λ链约510bp)的存在。
扩增子纯化、分析、定量、且制备用于454测序
为了消除PCR样品中的过量引物和/或引物二聚体,遵循制造商的方案(000387v001)使用Agentcourt Ampure磁珠(Beckman Coulter 目录号A63881)纯化扩增子。对于重链,合并所有四个反应(VH1/7、VH2/5/6、VH3、VH4),并且作为一份样品纯化,因此对于每个cDNA扩增纯化总共3份扩增子样品(重链、κ链和λ链)。用于ampure纯化的方案的修改在于纯化使用通用磁性机架在单一1.5 ml微管中进行,所述通用磁性机架适合用于1.5 ml管而非96孔板形式。所有体积和其他程序如方案中所述。然后通过遵循制造商的方案在Agilent 2100生物分析仪上使用高灵敏度DNA芯片(Agilent Technologies 目录号5067-4626)分析Ampure纯化后洗脱的扩增子的纯度和不存在任何污染的DNA种类。
一旦扩增子的纯度得到验证,则如制造商的方案中所述使用Quant-iT PicoGreen dsDNA测定试剂盒(Invitrogen 目录号P7589)在荧光计上定量DNA的浓度。试剂盒中提供的λ DNA用作浓度标准品,使用所述浓度标准品从100ng/孔至1.56ng/孔生成标准曲线。一式两份测定在TE缓冲液中100倍稀释的每种扩增子的荧光,且根据标准曲线的线性部分测定DNA的浓度。所有荧光测量在黑色96孔板中进行。如果荧光值在标准曲线的线性范围之外,则以更大或更小的稀释度对样品进行重新测量,以捕获落在线性范围内的荧光值。使用每种链类型的碱基对的近似大小(重-540bp,κ-485bp和λ-510bp),下式用来确定浓度:
每种扩增子的浓度(分子数/μl) =
[样品浓度(ng/μl)*6.022x1023]/[656.6x109*扩增子长度(bp)]
将每种扩增子归一化到1 X 107个分子/μl,然后以3:3:1(体积)的Hc:Kc:Lc的比例混合,涡旋,且最终1:10稀释,以获得1x106个分子/μl的最终浓度的混合物。
乳液PCR扩增、珠粒富集、珠粒计数和测序
遵循454公开的方案进行乳液PCR: "emPCR Amplification Method Manual – Lib-L" (Edition: May 2010 (Rev. April 2011)),其具有以下修改:
部分3.1.3 步骤2)
| 试剂 | 体积(μl) |
| 分子生物学级水 | 458 |
| 添加剂 | 515 |
| 扩增混合物 | 270 |
| 扩增引物 | 32 |
| 酶混合物 | 70 |
| PPiase | 2 |
| 总计 | 1347 |
一旦富集测序珠粒,根据"emPCR Amplification Method Manual – Lib-L"的步骤3.7,用以下设置在Beckman Coulter's Z2 Particle Counter上对珠粒计数:
孔径: <100 μm>
孔径 Kd: <60.04>
设置上截止值: <30.00μm>
设置下截止值: <10.00μm>
计数模式: <between>
定量体积: <0.5 ml>
分辨率: <256>。
珠粒的浓度计算为:
珠粒浓度= [来自颗粒计数的平均读数 * 4]珠粒/μl。
遵循454测序方案在454测序仪(Roche)上对从乳液PCR富集的珠粒进行测序:"Sequencing Method Manual – GS Junior Titanium Series" – May 2010 (Rev. June 2010),整体通过引用并入本文。
II. 肽片段的生成:
从人供体血浆纯化抗原特异性IgG
供体血浆分离和筛选对特异性抗原的反应性。
遵循IRB指南在肝素管中收集来自人志愿者的全血。在PBMC的聚蔗糖(ficoll)梯度分离(如上所述)过程中,同时收集血浆样品并且储存于-80℃。通过ELISA测试血浆IgG对各种抗原的反应性。简而言之,高结合的96孔板(Costar目录号)用100μl/孔的以2μg/ml溶解在碳酸盐缓冲液中的抗原在37℃下包被2小时或在4℃下包被过夜。将该板用PBS-Tween (0.1%)冲洗3次,然后用300μl/孔的PBS-Tween中的5%无脂干奶在37℃下封闭1小时。血浆样品以1/100、1/500和1/1000和1/2000稀释在5%奶PBS-Tween中,并且将100μl每种稀释物添加至96孔板中一式两份的经封闭的孔中,并且在37℃下 孵育2小时。将板用1xPBS-Tween洗涤3次,并且将以1/4000稀释在PBS-Tween中的辣根过氧化物酶缀合的抗人IgG抗体(Southern Biotech 2040-05)添加至各孔(100μl),并且在37℃下孵育一小时。该板用PBS-Tween洗涤6次,并且通过添加50μl TMB底物溶液(BioFX目录号TMBW-1000-01)和随后的50μ1终止溶液(BioFX目录号STPR1000-01)进行显色。测定在450nm处的光密度的信号。选择其血浆在1/500或更高稀释度显示显著信号的供体用于通过NG-XMT程序筛选。
乙型肝炎病毒小表面抗原(HBsAg) adw亚型购自Prospec (Rehovot, Israel,目录号HBS-872)。
从总血浆IgG纯化抗原特异性IgG
蛋白G纯化
1. 将5ml Protein G Sepharose 4 Fast Flow (GE Healthcare 目录号17-0618-05)的珠粒浆(2.5ml珠粒床体积)应用至重力流柱,并且用lxPBS洗涤两次。
2. 将用lxPBS稀释至15 ml的5ml人血浆应用至具有珠粒的柱上,并且该柱在旋转器上在4℃下孵育过夜,或室温下孵育2小时。
3. 该柱用20ml 1xPBS洗涤4次。
4. IgG用20ml pH2.7 0.1M甘氨酸/ HCl缓冲液洗脱并收集在含有用于中和的1.2ml 1M Tris pH8.5的管中。
5. 将10ml 1xPBS (pH7.4)添加至中和的洗脱物中,以使由于高浓度的IgG导致的沉淀最小化。
6. 纯化的IgG在10kDa截止透析盒(Pierce 目录号66456)中针对4升1xPBS透析两次。
7. 通过在Nanodrop分光光度计(Thermo Scientific)上测量280nm处的吸光度而测定IgG浓度。
亲和纯化
1. 遵循制造商的方案将HbsAg与生物素(Pierce 目录号20217)缀合。缀合的抗原在lxPBS中充分透析。
2. 2mg生物素缀合的抗原与5ml链霉抗生物素蛋白磁珠(Thermo Scientific目录号8816)在旋转器上在4℃下孵育过夜或在室温下孵育两个小时。珠粒用lxPBS冲洗两次,然后分到九个1.5 ml管中。
3. 抗原固定化至珠粒的效率通过HBsAg Elisa进行评价,并且一致显示大于80%的结合。
4. 向含有固定化的抗原的每个管中,添加来自单一供体的1mg蛋白G纯化的IgG,珠粒通过涡旋完全再悬浮,并且在室温下旋转孵育15 min。
5. 将试管放置在磁性机架中,去除上清液,珠粒用1ml lxPBS洗涤5次。
6. 最后洗涤步骤后,将0.9ml 0.1M甘氨酸-HCl缓冲液(pH1.8)应用至一管中,涡旋,并且在室温下孵育5 min。5分钟后,将第一管置于磁性机架上,然后去除管中的酸性缓冲液,并置于第二管中。重复该程序,直到所有9个试管培养用酸性缓冲液孵育。洗脱的IgG最后收集在含有用于中和的0.14ml 1M Tris pH8.5的管中。
7. 每管进行洗脱后,将珠粒用lxPBS洗涤两次,然后从其中将lmg蛋白G纯化的IgG添加至珠粒的步骤重新开始纯化。该程序重复多次,以便在MS分析之前生成用于蛋白酶处理的足够材料。
III. 质谱
如上所述进行质谱分析。简而言之,用蛋白酶(如胰蛋白酶)和/或化学蛋白切割试剂(例如溴化氰)消化之后,对肽进行质谱分析。将获得的MS2质谱与源自遗传物质数据库中的信息的理论MS2谱相关联,以鉴定编码特异性结合乙型肝炎病毒小表面抗原的抗体的基因序列。
IV. 单克隆抗体的表达和鉴定
以组合形式表达24个不同的重(γ)链可变区克隆、20个不同的κ链可变区克隆和10个不同的λ链可变区克隆,并且筛选抗原特异性结合活性(参见表14-15,其中γ链克隆在最左边的垂直列中注明,且轻链克隆在顶部水平行中注明)。每条γ链与每条轻(κ和λ)链配对,以通过在标准96孔组织培养板中瞬时转染HEK293E细胞而表达抗体。
通过酶联免疫吸附测定(ELISA)筛选从各孔中的转染细胞分泌的抗体与纯化的重组乙型肝炎表面抗原(购自Prospec, Ness-Ziona, ISRAEL的HBsAg-adw亚型)的结合。高结合的96孔ELISA板(Costar-3369)用50μl/孔的以2μg/ml稀释在碳酸盐缓冲液中的HBsAg通过在37℃下孵育2小时而包被,然后用300μl/孔的磷酸盐缓冲液(PBS)中的5%干乳通过在37℃下孵育1小时而封闭。来自瞬时转染的HEK293E细胞的上清液5倍稀释在含有0.05% Tween 20的PBS(PBS-T)中的5%乳中,然后将50μl稀释的上清液应用于HBsAg包被的ELISA板的各孔中。为了评价非特异性结合,还将相同上清液应用至仅用PBS中的5%乳包被的板。添加上清液后,ELISA板在37℃下孵育2小时,然后用250μl/孔的PBS-T洗涤3次。为了检测任何抗体结合,将4000倍稀释在PBS-T中的50μl/孔的辣根过氧化物酶(HRP)缀合的抗人IgG(Southern Biotech)添加至到各孔中,并且在37℃下孵育1小时。如上所述将板洗涤6次,然后添加50μl/孔HRP的生色底物3,3',5,5'-四甲基联苯胺,其约10分钟后用50μl/孔的酸进行中和。来自用酸中和的生色底物的信号通过450nm处的吸光度(光密度)进行测量。
表14-15显示从HBsAg板的吸光度减去各孔中只有乳的板的吸光度获得的值。以下上清液样品用作对照(值是在每种情况下两个独立孔的平均值): 阳性=来自抗HBsAg人抗体重链和轻链转染的上清液;阴性=来自仅用PEI转染的细胞的上清液。信号大于阴性对照信号10-20倍、20-40倍和高于40倍的孔用渐增的灰色阴影表示。30种重-轻排列表现出与HbsAg的强烈反应性,其在四个孔中的两个或更多个中是背景的大于40倍,26种是背景的20至40倍之间,18种是背景的10至20倍之间(18种的一种,表示为EVUGG γ链的组合,在表中用*显示,并且后来发现AKUOL λ链不反应)。因此,当与测试的30种轻链克隆中的至少一种配对时,在测试的24种不同的可变区γ链克隆中,17种表达HBsAg特异性抗体。
表 14
表 14续
表 14续
表 15
实施例9
在本实施例中,向人受试者施用包含目标抗原的疫苗,并且血液样品在接种之前(第0周)和然后在第1周和第2周进行采集。以4周间隔采集随后样品,最长达第52周。PBMC如实施例8中所述分离,或冻存在胎牛血清中的20% DMSO中或立即处理用于分离B细胞。血浆样品储存在-80℃,用于以后通过质谱分析。对于每份样品,PBMC和血浆如下所述进行处理,以评价接种后随时间的抗原特异性抗体群体。
I. 生成核酸序列
抗原特异性、记忆性和总B细胞分离和RNA纯化
为了分离B细胞,遵循制造商的方案使用Invitrogen's Dynabeads Untouched B-细胞分离试剂盒(Invitrogen 目录号113-51D)使用阴性选择方法来从PBMC消除所有非B细胞。将获得的未标记的B细胞群体进一步处理以分离抗原特异性或记忆性B细胞。
为了分离抗原特异性B细胞,在室温下在旋转器上将总的未标记的B细胞与固定在链霉抗生物素蛋白磁珠(Pierce-Thermo Scientific目录号88816)上的生物素化的抗原孵育20分钟。然后含有任何抗原结合的B细胞的珠粒用1xPBS洗涤两次。洗涤的珠粒然后重悬浮在Qiagen's RNeasy试剂盒RLT裂解缓冲液(补充有1%β-巯基乙醇)中,用于分离RNA。
为了分离记忆性B细胞,使用用于CD27+和表面IgG+细胞分离的Miltenyi's MACS试剂盒(Miltenyi Biotec (Auburn, CA) 目录号130-051-601和130-047-501)从总的未标记的B细胞分离CD27+和表面IgG+细胞。为了同时分离CD27+和sIgG+ B-细胞,在孵育步骤过程中同时添加针对这两种细胞表面标记的磁珠缀合的抗体。纯化后,将记忆性B细胞以300xg离心10分钟,然后如上对于RNA分离所述在用于RNA的RLT缓冲液中裂解。
遵循制造商的方案使用Qiagen's RNeasy试剂盒(Qiagen 目录号74104)从选择的细胞纯化RNA。通过引入DNase I消化步骤进行在柱上DNase I处理以消除污染的基因组DNA。RW1缓冲液洗涤后,将RDD缓冲液中稀释的DNase I (Qiagen 目录号79254)应用于RNA纯化柱,且在室温下孵育20分钟。然后将柱再一次用RW1缓冲液洗涤,随后用RPE缓冲液洗涤两次,且用30或50μ1水洗脱RNA。通过在Nanodrop分光光度计(Thermo Scientific)上在波长450nm处测量的吸光度测定测定RNA的浓度。
cDNA合成和通过PCR生成扩增子
首先如实施例8中所述逆转录从记忆性或抗原特异性B细胞分离的RNA。用于测序的重链、κ和λ链可变区的扩增子如下通过PCR生成。为了扩增重链,对于各cDNA样品使用实施例8中所述的引物运行4个独立的反应(每一个对于VH1和7;VH2、5和6;VH3;和VH4的基因家族是特异性的),以保持在B细胞合并物中VH基因转录物频率的自然分布。对于κ和λ链扩增,对于各cDNA样品运行每条链的单一反应。对于各反应,使用正向引物与相同浓度的反向引物的等摩尔混合物。使用对于Lib-L 平台的454测序(Roche)相容的融合引物进行扩增。反向引物设计为与每条链的恒定区的5'末端杂交。这些引物含有Lib-L引物B和MID序列,从而使得测序读取从各恒定区的最5'末端(以相反的意义)开始,且进入可变区的3'末端。对于重链和κ链,对于各MID使用单一反向引物,而对于λ链,对于各MID需要两条不同的反向引物。
PCR扩增使用Finnzyme's Phusion Hot Start II聚合酶(Thermo Scientific目录号F-540S)进行,其中反应混合物和条件如实施例8中所述设置。
为了确保不存在从任何试剂中的污染模板的任何虚假扩增,对于每种混合物设置一式两份的反应(对于重链4个独立的反应,对于每种轻链一个反应),其中用水取代cDNA模板。将这些无模板的阴性对照反应与含有模板的样品同时运行。完成PCR程序后,当将模板添加至反应但在cDNA存在的情况下,3μ1每个反应(包括阴性对照)在1.5%TAE琼脂糖凝胶上通过电泳分析扩增子(对于重链约540bp,对于κ链约485bp且对于λ链约510bp)的存在。
为了在测序过程中保留抗体链的同源配对,将分离的B细胞进行使用单细胞微滴封装法(Raindance Technologies, Inc., Lexington, MA)的单细胞封装。然后将封装的B细胞与单细胞RT-PCR试剂(由Qiagen作为目录号210210销售的试剂)以及扩增引物融合,以便从每个单个B细胞生成连接的重链和轻链PCR产物。重叠PCR(Meijer P.J.等人, J. Mol. Biol. 358(3):764-72, 2006)用于将重链和轻链PCR产物缝合为一个DNA,用于通过下游测序保存抗体链配对。
扩增子纯化、分析、定量、且制备用于454测序
为了消除PCR样品中的过量引物和/或引物二聚体,遵循制造商的方案(000387v001)使用Agentcourt Ampure磁珠(Beckman Coulter 目录号A63881)纯化扩增子。对于重链,合并所有四个反应(VH1/7、VH2/5/6、VH3、VH4),并且作为一份样品纯化,因此对于每个cDNA扩增纯化总共3份扩增子样品(重链、κ链和λ链)。用于Ampure纯化的方案的修改在于纯化使用通用磁性机架在单一1.5 ml微管中进行,所述通用磁性机架适合用于1.5 ml管而非96孔板形式。所有体积和其他程序如方案中所述。然后通过遵循制造商的方案在Agilent 2100生物分析仪上使用高灵敏度DNA芯片(Agilent Technologies 目录号5067-4626)分析Ampure纯化后洗脱的扩增子的纯度和不存在任何污染的DNA种类。
一旦扩增子的纯度得到验证,则如制造商的方案中所述使用Quant-iT PicoGreen dsDNA测定试剂盒(Invitrogen 目录号P7589)在荧光计上定量DNA的浓度。试剂盒中提供的λ DNA用作浓度标准品,使用所述浓度标准品从100 ng/孔至1.56 ng/孔生成标准曲线。一式两份测定在TE缓冲液中100倍稀释的每种扩增子的荧光,且根据标准曲线的线性部分测定DNA的浓度。所有荧光测量在黑色96孔板中进行。使用每种链类型的碱基对的近似大小(重-540bp,κ-485bp和λ-510bp),下式用来确定浓度:
每种扩增子的浓度(分子数/μl) =
[样品浓度(ng/μl)*6.022x1023]/[656.6x109*扩增子长度(bp)]
将每种扩增子归一化到1 X 107个分子/μl,然后以3:3:1(体积)的Hc:Kc:Lc的比例混合,涡旋,且最终1:10稀释,以获得1x106个分子/μl的最终浓度的混合物。
乳液PCR扩增、珠粒富集、珠粒计数和测序
遵循454公开的方案进行乳液PCR: "emPCR Amplification Method Manual – Lib-L" (Edition: May 2010 (Rev. April 2011)),其具有实施例8中所述的修改。
一旦富集测序珠粒,根据"emPCR Amplification Method Manual – Lib-L"的步骤3.7,在Beckman Coulter's Z2 Particle Counter上对珠粒计数,并且如下计算珠粒的浓度:
珠粒浓度= [来自颗粒计数的平均读数 * 4]珠粒/μl
遵循454测序方案在454测序仪(Roche)上对从乳液PCR富集的珠粒进行测序: "Sequencing Method Manual – GS Junior Titanium Series" – May 2010 (Rev. June 2010), 整体通过引用并入本文。
II. 肽片段的生成:
从人供体血浆纯化抗原特异性IgG
筛选对抗原的反应性。
通过ELISA测试血浆IgG对目标抗原的反应性。简而言之,高结合的96孔板(Costar目录号)用100μl/孔的以2μg/ml溶解在碳酸盐缓冲液中的抗原在37℃下包被2小时或在4℃下包被过夜。将该板用PBS-Tween (0.1%)冲洗3次,然后用300μl/孔的PBS-Tween中的5%无脂干奶在37℃下封闭1小时。血浆样品以1/100、1/500和1/1000和1/2000稀释在5%奶PBS-Tween中,并且将100μl每种稀释物添加至96孔板中一式两份的经封闭的孔中,并且在37℃下孵育2小时。将板用1x PBS-TWEEN洗涤3次,并且将以1/4000稀释在PBS-Tween中的辣根过氧化物酶缀合的抗人IgG抗体(Southern Biotech 2040-05)添加至各孔(100μl),并且在37℃下孵育一小时。该板用PBS-Tween洗涤6次,并且通过添加50μl TMB底物溶液(BioFX目录号TMBW-1000-01)和随后的50μ1终止溶液(BioFX目录号STPR1000-01)进行显色。测定在450nm处的光密度的信号。接种后,观察到血清滴度通常随时间推移而增加。
从总血浆IgG纯化抗原特异性IgG
如实施例8中所述使用蛋白G从每份血清样品纯化总IgG。纯化的IgG在10kDa截止透析盒(Pierce 目录号66456)中针对4升1xPBS透析两次, 并且通过在Nanodrop分光光度计(Thermo Scientific)上测量280nm处的吸光度而测定IgG浓度。然后如实施例8中所述使用结合抗原的珠粒亲和纯化蛋白G纯化的IgG。收集从每份样品中亲和纯化的抗体用于质谱分析。
III. 质谱
如上所述进行质谱分析。简而言之,用蛋白酶(如胰蛋白酶)和/或化学蛋白切割试剂(例如溴化氰)消化之后,对肽进行质谱分析。将获得的MS2质谱与源自遗传物质数据库中的信息的理论MS2谱相关联,以鉴定编码特异性结合目标抗原的抗体的基因序列。通过确定样品中的抗体序列,确定接种后多个时间点受试者中的抗原特异性抗体群体的组成。
实施例10
本实施例描述了使用表达人抗体基因的转基因动物来产生抗原特异性人抗体。
XENOMOUSE品系XMG1-KL小鼠(Amgen, Thousand Oaks, CA)的内源性小鼠抗体机制被失活,并且含有人免疫球蛋白重链和轻链基因座(Jakobovits等人, 2007, Nature Biotechnol., 25:1134-43)。这些小鼠产生完全人IgG1κ和IgG1λ抗体。用目标人抗原免疫小鼠,并且使用以下方法生成遗传物质数据库和肽数据库。
I. 遗传物质数据库:
细胞分离。
来自免疫小鼠的脾脏使用注射器和21G针以5 mL RPMI/10%FCS冲洗5次。将细胞冷冻在90% FCS/10% DMSO中。从每个脾分离总共50-100 x 10^6个细胞。
RNA分离和cDNA合成。
根据制造商的方案,使用QIAshredder (Qiagen目录号79654)和RNeasy微小试剂盒(Qiagen, Hilden, Germany;目录号74104)从脾细胞中分离总RNA。根据标准的下一代测序方案将RNA在柱上DNAse处理。使用ND-1000分光光度计(NanoDrop;由Thermo Scientific, Wilmington, DE商售) 测定总RNA浓度。
分离的RNA用于使用Thermoscript RT-PCR系统(Invitrogen (Life Technologies的部分), Carlsbad, CA目录号11146-024)通过逆转录合成第一链cDNA。根据制造商的方案使用1.5ug RNA和寡聚dT引物合成cDNA。
VH和VL扩增
用于测序的重链、κ和λ链可变区的扩增子如下使用如实施例8中所述的对人抗体序列特异性的引物通过PCR生成。为了扩增重链,对于各cDNA样品运行4个独立的反应(每一个对于VH1和7;VH2、5和6;VH3;和VH4的基因家族是特异性的),以保持在B细胞合并物中VH基因转录物频率的自然分布。对于κ和λ链扩增,对于各cDNA样品运行每条链的单一反应。对于各反应,使用正向引物与相同浓度的反向引物的等摩尔混合物。使用对于Lib-L 平台的454测序(Roche)相容的融合引物进行扩增。反向引物设计为与每条链的恒定区的5'末端杂交。这些引物含有Lib-L引物B和MID序列,从而使得测序读取从各恒定区的最5'末端(以相反的意义)开始,且进入可变区的3'末端。对于重链和κ链,对于各MID使用单一反向引物,而对于λ链,对于各MID需要两条不同的反向引物。
PCR扩增使用Finnzyme's Phusion Hot Start II聚合酶(Thermo Scientific目录号F-540S)进行,其中反应混合物和条件如实施例8中所述设置。
为了确保不存在从任何试剂中的污染模板的任何虚假扩增,对于每种混合物设置一式两份的反应(对于重链4个独立的反应,对于每种轻链一个反应),其中用水取代cDNA模板。将这些无模板的阴性对照反应与含有模板的样品同时运行。完成PCR程序后,当将模板添加至反应但在cDNA存在的情况下,3μ1每个反应(包括阴性对照)在1.5%TAE琼脂糖凝胶上通过电泳分析扩增子(对于重链约540bp,对于κ链约485bp且对于λ链约510bp)的存在。根据制造商的方案,使用AMPure XP (Agencourt; Beckman Coulter Genomics, Brea, CA, 目录号A63881)纯化PCR产物,且使用Agilent 2100 BioAnalyzer进行分析。
然后将PCR产物的序列翻译成预测的氨基酸序列,然后将所述预测的氨基酸序列理论消化(例如,用蛋白酶和/或化学蛋白切割试剂),以产生虚拟的肽片段。然后这些虚拟的肽片段用于生成预测质谱。
从多克隆抗体的肽片段生成实际质谱:
从小鼠的血清和/或血浆纯化多克隆抗体。为了纯化抗体,使用以下方法:
蛋白-G纯化:
将1mL蛋白-G磁珠(Millipore (Billerica, MA), 目录号LSKMAGG10)添加到四个15 ml锥形管(Falcon (BD Biosciences, Franklin Lake, NJ),目录号352097)中的每一个中。每个管中的珠粒用10mL磷酸盐缓冲盐水pH7.4, 0.05% Tween-20 (PBST)洗涤两次,且用10mL PBS洗涤三次。将来自三只小鼠(ID 1262-2, 1262-4, 1263-4)的血清合并在一起,在PBS中稀释10倍至6ml终体积。然后将1.5ml合并且稀释的血清添加至每个试管中的珠粒,且在4℃下孵育过夜。将流通物收集,且再投入通过纯化过程两次。收集流通物后,每管用10mL PBST洗涤两次,且用10mL PBS洗涤三次。然后每管在4℃下用0.5mL 0.1M pH 2.7甘氨酸孵育30分钟以洗脱IgG。洗脱重复5次。所有洗脱物用1M Tris pH 8.5中和,针对PBS透析过夜,且用ND-1000分光光度计(Nanodrop)测定蛋白浓度。总共纯化约2.5mg IgG。
抗原柱制备:
5.0mL新鲜的链霉抗生物素蛋白(SA)磁珠(Pierce,目录号88817)用10mL PBS洗涤三次,且在4℃下与稀释在5.0 mL PBS中的105 uL 20mg/ml缀合至生物素的目标抗原的储存物孵育过夜。弃去流通物,珠粒用10mL PBS洗涤三次,且等分至10个低结合1.7mL管(Axygen (Union City, CA), 目录号MCT-175-L-C)。将等分的珠粒置于磁性机架(Invitrogen, DynaMag)上,去除PBS,然后添加稀释的血清。
抗原特异性纯化:
将来自上述的蛋白-G纯化的IgG添加至与生物素化的抗原偶联的SA-磁珠。在4℃下过夜孵育后,收集流通物,且连续地用总共10mL以下各缓冲液洗涤珠粒:
PBS
RIPA缓冲液(即放射免疫测定缓冲液;Alcaraz 等人, J. Vet. Diagn. Invest. 2(3): 191-196, 1990; Ngoka, L.C., Proteome Sci. 6(1): 30, 2008)
PBS 中20%乙腈
PBS 中60%乙二醇
PBS 中0.5M NaCl
PBS (即磷酸盐缓冲盐水)
IgG然后用5份的1.5mL 0.1M甘氨酸pH 3.5洗脱,然后用5份的1.5mL 0.1M甘氨酸pH 2.7洗脱,然后用5份的1.5mL 0.1M甘氨酸pH 1.8洗脱,且用1M Tris pH 8.5中和。使用以抗原包被的96孔板针对与目标抗原的反应性测定洗脱物。具有活性的级分通过ELISA (Thermo, 目录号23300)进行定量,并且通过western印迹测定抗原反应性。具有最干净的反应性的级分通过质谱分析。
质谱
如上所述进行质谱分析。简而言之,用蛋白酶(如胰蛋白酶)和/或化学蛋白切割试剂(例如溴化氰)消化之后,对肽进行质谱分析。将获得的MS2谱与源自遗传物质数据库中的信息的理论MS2谱相关联,以鉴定编码特异性结合目标抗原的抗体的基因序列。
单克隆抗体的表达和鉴定
不同的重链(γ)可变区克隆、κ链可变区克隆和λ链可变区克隆以组合形式进行表达,并且筛选抗原特异性结合活性。每条γ链与每条轻(κ和λ)链配对,以通过在标准96孔组织培养板中瞬时转染HEK293E细胞而表达抗体。
如上所述,通过酶联免疫吸附测定(ELISA)筛选从各孔中的转染细胞分泌的抗体与纯化的重组抗原的结合。重链和轻链的几种配对导致产生特异性结合抗原包被板的抗体。选择这些重链和轻链对,导致产生特异性结合目标人抗原的完全人抗体。
等同内容
仅仅使用常规实验,本领域技术人员将认识到或者能够确定本文具体描述的实施方案的许多等同内容。下列权利要求的范围旨在涵盖这些等同内容。
Claims (50)
1.用于获得特异性结合抗原的免疫球蛋白的重链或轻链的核苷酸序列或氨基酸序列的方法,其包括:
(a) 提供编码至少一种动物的免疫球蛋白链或其可变区的核酸序列;
(b) 获得特异性结合抗原的多克隆免疫球蛋白群体,并且获得所述群体的重链或轻链或其可变区的肽片段的质谱信息;
(c) 将所述肽片段的质谱信息与从所述核酸序列预测的质谱信息相关联,以鉴定包含所述肽片段的免疫球蛋白链(或其可变区)的核苷酸序列或氨基酸序列,其中所述预测质谱信息源自所述核酸序列编码的预测氨基酸序列;和
(d) 基于所述肽片段对所述免疫球蛋白链或其片段的氨基酸序列覆盖率从鉴定的免疫球蛋白链(或其可变区)的核苷酸序列或氨基酸序列中选择,以获得特异性结合抗原的免疫球蛋白的重链或轻链的核苷酸序列或氨基酸序列。
2.权利要求1的方法,其中所述步骤(a)的至少一种动物是暴露于所述抗原的动物。
3.权利要求1的方法,其中所述核酸序列是表达的核酸序列。
4.权利要求1的方法,其中所述编码免疫球蛋白链(或其可变区)的核酸序列通过以下从所述至少一种动物获得:
(1) 从来自所述至少一种动物的白血细胞分离核酸分子;和
(2) 使用对于在编码所述免疫球蛋白链(或其可变区)的核酸分子邻近的多核苷酸序列特异性的引物扩增编码所述免疫球蛋白链(或其可变区)的核酸分子;和
(3) 获得编码免疫球蛋白链(或其可变区)的所述扩增的核酸分子的核苷酸序列。
5.权利要求4的方法,其中编码多肽的核酸分子是RNA分子,并且所述扩增步骤包括初始的逆转录步骤。
6.权利要求4的方法,其中所述与编码所述免疫球蛋白链(或其可变区)的核酸分子邻近的多核苷酸序列选自免疫球蛋白基因侧翼的基因组DNA、编码免疫球蛋白链恒定区的多核苷酸序列、和编码免疫球蛋白链构架区的多核苷酸序列。
7.权利要求1的方法,其中所述预测的质谱信息使用包括以下步骤的方法来获得:
(i) 用一种或多种蛋白酶和/或一种或多种化学蛋白切割试剂进行所述核酸序列的核苷酸序列编码的预测氨基酸序列的理论消化来生成虚拟的肽片段;和
(ii) 生成所述虚拟肽片段的预测质谱。
8.权利要求1的方法,其中所述步骤(a)的核酸序列、预测的氨基酸序列和源自所述核酸序列的预测质谱位于遗传物质数据库中。
9.权利要求1的方法,其中所述步骤(b)的所述多克隆免疫球蛋白群体从动物的体液样品或其部分获得。
10.权利要求9的方法,其中所述体液选自血液、脑脊液、滑液、腹膜液、粘膜分泌物、泪液、鼻分泌物、唾液、乳和泌尿生殖道分泌物。
11.权利要求9的方法,其中所述动物是以前暴露于所述抗原的动物。
12.权利要求11的方法,其中所述以前暴露于所述抗原的动物是以前用所述抗原免疫的动物。
13.权利要求9的方法,其中所述动物与步骤(a)中的所述至少一种动物是相同的。
14.权利要求9的方法,其中所述步骤(b)的多克隆免疫球蛋白群体从所述体液样品或其部分纯化。
15.权利要求14的方法,所述纯化通过蛋白A或蛋白G纯化或基于所述抗原的亲和纯化来实现。
16.权利要求1的方法,其中所述步骤(b)的多克隆免疫球蛋白群体从体外培养细胞的培养基中获得。
17.权利要求1的方法,其中所述步骤(b)的肽片段的质谱信息通过用一种或多种蛋白酶和/或一种或多种化学蛋白切割试剂消化所述群体以生成片段并且使生成的片段进行质谱分析而从所述多克隆免疫球蛋白群体获得。
18.权利要求1的方法,其中所述步骤(d)中的选择基于所述肽片段对所述免疫球蛋白的CDR3序列的氨基酸序列覆盖率。
19.权利要求1的方法,其中所述步骤(d)中的选择基于所述肽片段对所述免疫球蛋白的可变区的氨基酸序列覆盖率。
20.权利要求1的方法,其中所述步骤(d)中的选择基于所述肽片段对所述免疫球蛋白的CDR3序列的氨基酸序列覆盖率和可变区的氨基酸序列覆盖率。
21.权利要求1的方法,其中所述步骤(d)中的选择另外基于选自以下的至少一种参数:映射的独特肽的数量、谱共享、总肽计数、独特肽计数、编码核酸序列的频率和克隆相关性。
22.生成特异性结合抗原的免疫球蛋白的方法,其包括:
通过使用权利要求1的方法获得重链和轻链的核苷酸序列或氨基酸序列,
通过重组分子生物学技术或基因合成技术基于获得的核苷酸序列或氨基酸序列制备所述重链和所述轻链,和
将所述重链与所述轻链装配以生成特异性结合所述抗原的免疫球蛋白。
23.权利要求22的方法,其进一步包括在免疫测定中评价所述免疫球蛋白以确认所述免疫球蛋白特异性结合所述抗原。
24.权利要求23的方法,其中所述免疫测定选自流式细胞术测定、酶联免疫吸附测定(ELISA)、Western印迹测定、免疫组织化学测定、免疫荧光测定、放射免疫测定、中和测定、结合测定、亲和力测定、蛋白免疫沉淀测定和肽免疫沉淀测定。
25.用于获得特异性结合抗原的免疫球蛋白的免疫球蛋白链可变区的核苷酸序列或氨基酸序列的方法,其包括:
(a) 提供编码至少一种动物的多种免疫球蛋白的免疫球蛋白可变区的核酸序列;
(b) 获得特异性结合抗原的多克隆免疫球蛋白群体的免疫球蛋白链可变区的肽片段的质谱信息;
(c) 将所述肽片段的质谱信息与所述核酸序列的预测质谱信息相关联,以鉴定包含所述肽片段的免疫球蛋白链可变区的核苷酸序列或氨基酸序列,其中所述预测质谱信息源自所述核酸序列编码的预测氨基酸序列;和
(d) 基于所述肽片段对所述可变区的氨基酸序列覆盖率从鉴定的免疫球蛋白链可变区的核苷酸序列或氨基酸序列中选择,以获得特异性结合抗原的免疫球蛋白的可变区的核苷酸序列或氨基酸序列。
26.权利要求25的方法,其进一步包括
(e) 通过重组分子生物学技术或基因合成技术基于步骤(d)中获得的核苷酸序列或氨基酸序列制备所述免疫球蛋白链可变区。
27.权利要求26的方法,其进一步包括
(f) 用免疫测定筛选所述免疫球蛋白链可变区,以分离特异性结合所述抗原的免疫球蛋白的免疫球蛋白链可变区。
28.权利要求25的方法,其中所述免疫球蛋白链是重链。
29.权利要求25的方法,其中所述免疫球蛋白链是轻链。
30.用于生成特异性结合抗原的免疫球蛋白的抗原结合结构域的方法,其包括:
(a) 根据权利要求25的方法获得特异性结合抗原的免疫球蛋白的免疫球蛋白链可变区的核苷酸序列或氨基酸序列;和
(b) 将免疫球蛋白重链可变区与免疫球蛋白轻链可变区装配以生成特异性结合所述抗原的免疫球蛋白的抗原结合结构域。
31.权利要求1、22、25或30的方法,其中所述动物是人。
32.权利要求1、22、25或30的方法,其中所述动物是兔或小鼠。
33.根据权利要求22的方法生成的特异性结合抗原的免疫球蛋白。
34.根据权利要求25的方法分离的特异性结合抗原的免疫球蛋白的免疫球蛋白链可变区。
35.根据权利要求30的方法生成的特异性结合抗原的免疫球蛋白的抗原结合结构域。
36.包含权利要求33的免疫球蛋白和药学可接受的载体的组合物。
37.包含权利要求34的免疫球蛋白链可变区和药学可接受的载体的组合物。
38.包含权利要求35的抗原结合结构域和药学可接受的载体的组合物。
39.治疗患有或疑似患有特征在于疾病抗原的疾病的动物的方法,其中所述方法包括施用有效量的权利要求36、37或38的组合物,其中所述抗原和所述疾病抗原是相同的。
40.权利要求39的方法,其中所述动物是人。
41.降低动物中特征在于所述动物中存在疾病抗原的疾病的发生可能性的方法,其中所述方法包括施用有效量的权利要求36、37或38的组合物,其中所述抗原和所述疾病抗原是相同的。
42.权利要求41的方法,其中所述组合物进一步包含佐剂。
43.权利要求41的方法,其中所述动物是人。
44.监测暴露于抗原的受试者的抗原特异性抗体群体的方法,其包括:
(a) 获得编码来自暴露于抗原的受试者的多种免疫球蛋白的免疫球蛋白链(或其可变区)的核酸序列;
(b) 获得来自所述受试者的特异性结合所述抗原的多克隆免疫球蛋白群体的免疫球蛋白重链和免疫球蛋白轻链的肽片段的质谱信息;
(c) 将所述肽片段的质谱信息与所述核酸序列的预测质谱信息相关联,以鉴定来自所述受试者的特异性结合所述抗原的多克隆免疫球蛋白群体的序列,其中所述预测质谱信息源自所述核酸序列编码的预测氨基酸序列;和
(d) 基于肽片段对所述免疫球蛋白链或其片段的氨基酸序列覆盖率从鉴定的免疫球蛋白链(或其可变区)的核苷酸序列或氨基酸序列中选择,以获得特异性结合所述抗原的免疫球蛋白的重链或轻链的核苷酸序列或氨基酸序列。
45.权利要求44的方法,其中所述核酸序列和多克隆免疫球蛋白群体各自对应于所述受试者暴露于所述抗原之后的一个时间点。
46.权利要求44的方法,其中所述核酸序列和多克隆免疫球蛋白群体各自对应于所述受试者暴露于所述抗原之后的多个时间点。
47.权利要求33的免疫球蛋白用于制备药物的用途,所述药物可用于治疗或降低动物中特征在于疾病抗原的疾病的可能性,其中所述抗原和所述疾病抗原是相同的。
48.权利要求34的免疫球蛋白链可变区用于制备药物的用途,所述药物可用于治疗或降低动物中特征在于疾病抗原的疾病的可能性,其中所述抗原和所述疾病抗原是相同的。
49.权利要求35的抗原结合结构域用于制备药物的用途,所述药物可用于治疗或降低动物中特征在于疾病抗原的疾病的可能性,其中所述抗原和所述疾病抗原是相同的。
50.权利要求47-49中任一项的用途,其中所述动物是人。
Priority Applications (1)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| CN201810154961.6A CN108220297A (zh) | 2011-03-09 | 2012-03-09 | 用于生成单克隆抗体的方法和试剂 |
Applications Claiming Priority (9)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201161450922P | 2011-03-09 | 2011-03-09 | |
| US61/450,922 | 2011-03-09 | ||
| US201161560006P | 2011-11-15 | 2011-11-15 | |
| US61/560,006 | 2011-11-15 | ||
| US201161566876P | 2011-12-05 | 2011-12-05 | |
| US61/566,876 | 2011-12-05 | ||
| US201261594729P | 2012-02-03 | 2012-02-03 | |
| US61/594,729 | 2012-02-03 | ||
| PCT/US2012/028501 WO2012122484A1 (en) | 2011-03-09 | 2012-03-09 | Methods and reagents for creating monoclonal antibodies |
Related Child Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| CN201810154961.6A Division CN108220297A (zh) | 2011-03-09 | 2012-03-09 | 用于生成单克隆抗体的方法和试剂 |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| CN103842379A true CN103842379A (zh) | 2014-06-04 |
Family
ID=45922811
Family Applications (2)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| CN201810154961.6A Pending CN108220297A (zh) | 2011-03-09 | 2012-03-09 | 用于生成单克隆抗体的方法和试剂 |
| CN201280021326.1A Pending CN103842379A (zh) | 2011-03-09 | 2012-03-09 | 用于生成单克隆抗体的方法和试剂 |
Family Applications Before (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| CN201810154961.6A Pending CN108220297A (zh) | 2011-03-09 | 2012-03-09 | 用于生成单克隆抗体的方法和试剂 |
Country Status (12)
| Country | Link |
|---|---|
| US (2) | US9920110B2 (zh) |
| EP (2) | EP2683736B1 (zh) |
| JP (3) | JP6211931B2 (zh) |
| KR (3) | KR20190107761A (zh) |
| CN (2) | CN108220297A (zh) |
| AU (3) | AU2012225310B2 (zh) |
| CA (1) | CA2829606C (zh) |
| DK (1) | DK2683736T3 (zh) |
| ES (1) | ES2666301T3 (zh) |
| HK (1) | HK1256304A1 (zh) |
| HU (1) | HUE037095T2 (zh) |
| WO (1) | WO2012122484A1 (zh) |
Cited By (13)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN104560980A (zh) * | 2015-01-20 | 2015-04-29 | 中国人民解放军第三军医大学 | 基于高通量测序构建鼠BCR轻链Lamda文库的多重PCR引物和方法 |
| CN104560979A (zh) * | 2015-01-20 | 2015-04-29 | 中国人民解放军第三军医大学 | 基于高通量测序构建鼠bcr重链文库的多重pcr引物和方法 |
| CN105277638A (zh) * | 2014-07-22 | 2016-01-27 | 沈鹤霄 | 一种单克隆抗体IgG质谱测序方法 |
| CN106047857A (zh) * | 2016-06-01 | 2016-10-26 | 苏州金唯智生物科技有限公司 | 一种发掘特异性功能抗体的方法 |
| CN107525934A (zh) * | 2016-06-21 | 2017-12-29 | 张效龙 | 一种测定血液中抗体氨基酸序列的方法 |
| CN108348167A (zh) * | 2015-09-09 | 2018-07-31 | 优比欧迈公司 | 用于脑-颅面健康相关病症的源自微生物群系的诊断及治疗方法和系统 |
| CN108530533A (zh) * | 2018-04-13 | 2018-09-14 | 吉林大学 | 猪瘟病毒单克隆抗体hk24及医用用途 |
| CN109425662A (zh) * | 2017-08-23 | 2019-03-05 | 深圳华大基因研究院 | 一种鉴定蛋白的方法及系统 |
| CN110382696A (zh) * | 2017-03-07 | 2019-10-25 | 豪夫迈·罗氏有限公司 | 发现替代性抗原特异性抗体变体的方法 |
| CN110452296A (zh) * | 2019-07-16 | 2019-11-15 | 福州精锐生物技术有限公司 | 一种兔单克隆抗体制备方法 |
| CN111201238A (zh) * | 2017-07-21 | 2020-05-26 | 诺夫免疫股份有限公司 | 产生多特异性抗体混合物及其使用方法 |
| WO2023184734A1 (zh) * | 2022-03-31 | 2023-10-05 | 台州恩泽医疗中心(集团) | 一种抗hla-g1,-g2,-g5及hla-g6异构体分子的单克隆抗体及其用途 |
| CN110382696B (zh) * | 2017-03-07 | 2024-04-19 | 豪夫迈·罗氏有限公司 | 发现替代性抗原特异性抗体变体的方法 |
Families Citing this family (34)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| ES2726702T3 (es) | 2009-01-15 | 2019-10-08 | Adaptive Biotechnologies Corp | Perfilado de la inmunidad adaptativa y métodos para la generación de anticuerpos monoclonales |
| EP2783214A2 (en) | 2011-11-23 | 2014-10-01 | The Board of Regents of The University of Texas System | Proteomic identification of antibodies |
| EP2823060B1 (en) | 2012-03-05 | 2018-02-14 | Adaptive Biotechnologies Corporation | Determining paired immune receptor chains from frequency matched subunits |
| SG11201407107VA (en) | 2012-05-08 | 2014-11-27 | Adaptive Biotechnologies Corp | Compositions and method for measuring and calibrating amplification bias in multiplexed pcr reactions |
| CA2875695C (en) | 2012-06-15 | 2022-11-15 | The Board Of Regents Of The University Of Texas System | High throughput sequencing of multiple transcripts of a single cell |
| SG10201707394PA (en) * | 2013-03-15 | 2017-10-30 | Adaptive Biotechnologies Corp | Uniquely tagged rearranged adaptive immune receptor genes in a complex gene set |
| US9708657B2 (en) | 2013-07-01 | 2017-07-18 | Adaptive Biotechnologies Corp. | Method for generating clonotype profiles using sequence tags |
| US10208125B2 (en) | 2013-07-15 | 2019-02-19 | University of Pittsburgh—of the Commonwealth System of Higher Education | Anti-mucin 1 binding agents and uses thereof |
| CA2922372C (en) * | 2013-09-09 | 2018-05-15 | Shimadzu Corporation | Method for preparing peptide fragments, kit for preparing peptide fragments to be used therein, and analysis method |
| GB201316644D0 (en) * | 2013-09-19 | 2013-11-06 | Kymab Ltd | Expression vector production & High-Throughput cell screening |
| US10066265B2 (en) | 2014-04-01 | 2018-09-04 | Adaptive Biotechnologies Corp. | Determining antigen-specific t-cells |
| ES2777529T3 (es) | 2014-04-17 | 2020-08-05 | Adaptive Biotechnologies Corp | Cuantificación de genomas de células inmunitarias adaptativas en una mezcla compleja de células |
| EP3161166A4 (en) * | 2014-06-25 | 2017-11-29 | The Rockefeller University | Compositions and methods for rapid production of versatile nanobody repertoires |
| EP3212790B1 (en) | 2014-10-29 | 2020-03-25 | Adaptive Biotechnologies Corp. | Highly-multiplexed simultaneous detection of nucleic acids encoding paired adaptive immune receptor heterodimers from many samples |
| US10246701B2 (en) | 2014-11-14 | 2019-04-02 | Adaptive Biotechnologies Corp. | Multiplexed digital quantitation of rearranged lymphoid receptors in a complex mixture |
| AU2016222788B2 (en) | 2015-02-24 | 2022-03-31 | Adaptive Biotechnologies Corp. | Methods for diagnosing infectious disease and determining HLA status using immune repertoire sequencing |
| CN107635478A (zh) | 2015-03-06 | 2018-01-26 | 英国质谱公司 | 通过质谱或离子迁移谱进行的组织分析 |
| US11367605B2 (en) | 2015-03-06 | 2022-06-21 | Micromass Uk Limited | Ambient ionization mass spectrometry imaging platform for direct mapping from bulk tissue |
| GB2554206B (en) | 2015-03-06 | 2021-03-24 | Micromass Ltd | Spectrometric analysis of microbes |
| EP3264989B1 (en) | 2015-03-06 | 2023-12-20 | Micromass UK Limited | Spectrometric analysis |
| WO2016142693A1 (en) | 2015-03-06 | 2016-09-15 | Micromass Uk Limited | In vivo endoscopic tissue identification tool |
| CN110057901B (zh) * | 2015-03-06 | 2022-07-01 | 英国质谱公司 | 拭子和活检样品的快速蒸发电离质谱和解吸电喷雾电离质谱分析 |
| WO2016142674A1 (en) | 2015-03-06 | 2016-09-15 | Micromass Uk Limited | Cell population analysis |
| US10513733B2 (en) | 2015-03-23 | 2019-12-24 | Board Of Regents, The University Of Texas System | High throughout sequencing of paired VH and VL transcripts from B cells secreting antigen-specific antibodies |
| WO2016161273A1 (en) | 2015-04-01 | 2016-10-06 | Adaptive Biotechnologies Corp. | Method of identifying human compatible t cell receptors specific for an antigenic target |
| EP3443354A1 (en) | 2016-04-14 | 2019-02-20 | Micromass UK Limited | Spectrometric analysis of plants |
| US10428325B1 (en) | 2016-09-21 | 2019-10-01 | Adaptive Biotechnologies Corporation | Identification of antigen-specific B cell receptors |
| WO2019084538A1 (en) | 2017-10-27 | 2019-05-02 | Board Of Regents, The University Of Texas System | TUMOR SPECIFIC ANTIBODIES, T CELL RECEPTORS AND METHODS OF IDENTIFICATION THEREOF |
| US11254980B1 (en) | 2017-11-29 | 2022-02-22 | Adaptive Biotechnologies Corporation | Methods of profiling targeted polynucleotides while mitigating sequencing depth requirements |
| US11155575B2 (en) | 2018-03-21 | 2021-10-26 | Waters Technologies Corporation | Non-antibody high-affinity-based sample preparation, sorbent, devices and methods |
| CN108713136B (zh) * | 2018-03-29 | 2020-10-09 | 深圳达闼科技控股有限公司 | 物质检测方法、装置、电子设备及计算机可读存储介质 |
| US20220034902A1 (en) * | 2018-12-11 | 2022-02-03 | Merck Sharp & Dohme Corp. | Method for identifying high affinity monoclonal antibody heavy and light chain pairs from high throughput screens of b-cell and hybridoma libraries |
| WO2022216795A1 (en) * | 2021-04-09 | 2022-10-13 | Abterra Biosciences, Inc. | Method for antibody identification from protein mixtures |
| IT202100019334A1 (it) * | 2021-07-21 | 2023-01-21 | Univ Degli Studi Di Pavia | Metodo per l’identificazione dell’intera sequenza della regione variabile delle catene pesanti e leggere di immunoglobuline |
Citations (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN101679513A (zh) * | 2007-03-06 | 2010-03-24 | 西福根有限公司 | 用于治疗呼吸道合胞病毒感染的重组抗体 |
Family Cites Families (62)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US4485045A (en) | 1981-07-06 | 1984-11-27 | Research Corporation | Synthetic phosphatidyl cholines useful in forming liposomes |
| GB8308235D0 (en) | 1983-03-25 | 1983-05-05 | Celltech Ltd | Polypeptides |
| US4816567A (en) | 1983-04-08 | 1989-03-28 | Genentech, Inc. | Recombinant immunoglobin preparations |
| US4544545A (en) | 1983-06-20 | 1985-10-01 | Trustees University Of Massachusetts | Liposomes containing modified cholesterol for organ targeting |
| US6548640B1 (en) | 1986-03-27 | 2003-04-15 | Btg International Limited | Altered antibodies |
| US5075109A (en) | 1986-10-24 | 1991-12-24 | Southern Research Institute | Method of potentiating an immune response |
| US4897268A (en) | 1987-08-03 | 1990-01-30 | Southern Research Institute | Drug delivery system and method of making the same |
| US5530101A (en) | 1988-12-28 | 1996-06-25 | Protein Design Labs, Inc. | Humanized immunoglobulins |
| US6673776B1 (en) | 1989-03-21 | 2004-01-06 | Vical Incorporated | Expression of exogenous polynucleotide sequences in a vertebrate, mammal, fish, bird or human |
| US5013556A (en) | 1989-10-20 | 1991-05-07 | Liposome Technology, Inc. | Liposomes with enhanced circulation time |
| EP1690935A3 (en) | 1990-01-12 | 2008-07-30 | Abgenix, Inc. | Generation of xenogeneic antibodies |
| US6150584A (en) | 1990-01-12 | 2000-11-21 | Abgenix, Inc. | Human antibodies derived from immunized xenomice |
| US5427908A (en) | 1990-05-01 | 1995-06-27 | Affymax Technologies N.V. | Recombinant library screening methods |
| GB9015198D0 (en) | 1990-07-10 | 1990-08-29 | Brien Caroline J O | Binding substance |
| US5545806A (en) | 1990-08-29 | 1996-08-13 | Genpharm International, Inc. | Ransgenic non-human animals for producing heterologous antibodies |
| JPH07503132A (ja) | 1991-12-17 | 1995-04-06 | ジェンファーム インターナショナル,インコーポレイティド | 異種抗体を産生することができるトランスジェニック非ヒト動物 |
| NZ261259A (en) | 1993-01-12 | 1996-12-20 | Biogen Inc | Humanised recombinant anti-vla4 antibody and diagnostic compositions and medicaments |
| US6436908B1 (en) | 1995-05-30 | 2002-08-20 | Duke University | Use of exogenous β-adrenergic receptor and β-adrenergic receptor kinase gene constructs to enhance myocardial function |
| US5641870A (en) | 1995-04-20 | 1997-06-24 | Genentech, Inc. | Low pH hydrophobic interaction chromatography for antibody purification |
| US5739277A (en) | 1995-04-14 | 1998-04-14 | Genentech Inc. | Altered polypeptides with increased half-life |
| US5750341A (en) | 1995-04-17 | 1998-05-12 | Lynx Therapeutics, Inc. | DNA sequencing by parallel oligonucleotide extensions |
| US6376471B1 (en) | 1997-10-10 | 2002-04-23 | Johns Hopkins University | Gene delivery compositions and methods |
| US6780591B2 (en) | 1998-05-01 | 2004-08-24 | Arizona Board Of Regents | Method of determining the nucleotide sequence of oligonucleotides and DNA molecules |
| EP1082458A1 (en) | 1998-05-01 | 2001-03-14 | Arizona Board Of Regents | Method of determining the nucleotide sequence of oligonucleotides and dna molecules |
| US7300753B2 (en) | 1998-09-04 | 2007-11-27 | John Rush | Immunoaffinity isolation of modified peptides from complex mixtures |
| AR021833A1 (es) | 1998-09-30 | 2002-08-07 | Applied Research Systems | Metodos de amplificacion y secuenciacion de acido nucleico |
| US7244559B2 (en) | 1999-09-16 | 2007-07-17 | 454 Life Sciences Corporation | Method of sequencing a nucleic acid |
| US7211390B2 (en) | 1999-09-16 | 2007-05-01 | 454 Life Sciences Corporation | Method of sequencing a nucleic acid |
| AU2002323139A1 (en) | 2001-08-14 | 2003-03-03 | President And Fellows Of Harvard College | Absolute quantification of proteins and modified forms thereof by multistage mass spectrometry |
| CN1302006C (zh) * | 2001-12-14 | 2007-02-28 | 伊莱利利公司 | 重组牛凝血酶 |
| GB0130267D0 (en) | 2001-12-19 | 2002-02-06 | Neutec Pharma Plc | Focussed antibody technology |
| US7498024B2 (en) | 2003-06-03 | 2009-03-03 | Cell Genesys, Inc. | Compositions and methods for enhanced expression of immunoglobulins from a single vector using a peptide cleavage site |
| US7485291B2 (en) | 2003-06-03 | 2009-02-03 | Cell Genesys, Inc. | Compositions and methods for generating multiple polypeptides from a single vector using a virus derived peptide cleavage site, and uses thereof |
| US7169560B2 (en) | 2003-11-12 | 2007-01-30 | Helicos Biosciences Corporation | Short cycle methods for sequencing polynucleotides |
| EP1714152A4 (en) | 2004-02-09 | 2008-08-13 | Univ Northeastern | Monoclonal antibody-based biomarker detection and development platform |
| JP2007526772A (ja) | 2004-02-27 | 2007-09-20 | プレジデント・アンド・フェロウズ・オブ・ハーバード・カレッジ | インサイチュー配列決定用ポロニー蛍光ビーズ |
| WO2005084134A2 (en) | 2004-03-04 | 2005-09-15 | Dena Leshkowitz | Quantifying and profiling antibody and t cell receptor gene expression |
| PA8672101A1 (es) | 2005-04-29 | 2006-12-07 | Centocor Inc | Anticuerpos anti-il-6, composiciones, métodos y usos |
| CA2610804C (en) * | 2005-06-10 | 2013-11-19 | Ares Trading S.A. | Process for the purification of il-18 binding protein |
| TWI388568B (zh) | 2006-02-10 | 2013-03-11 | Genentech Inc | 抗fgf19抗體及其使用方法 |
| GB0612928D0 (en) | 2006-06-29 | 2006-08-09 | Ucb Sa | Biological products |
| CA2657223A1 (en) * | 2006-07-07 | 2008-01-10 | Pall Corporation | Use of denaturing agents during affinity capture |
| WO2008079914A1 (en) | 2006-12-21 | 2008-07-03 | Novartis Ag | Antibody quantitation |
| PL2132229T3 (pl) | 2007-03-01 | 2016-12-30 | Kompozycje rekombinowanych przeciwciał anty-receptor czynnika wzrostu naskórka | |
| KR101605908B1 (ko) | 2007-03-22 | 2016-03-23 | 바이오젠 엠에이 인코포레이티드 | Cd154에 특이적으로 결합하는, 항체, 항체 유도체 및 항체 단편을 포함하는 결합 단백질, 및 그의 용도 |
| PT2602323T (pt) | 2007-06-01 | 2018-04-02 | Open Monoclonal Tech Inc | Composições e métodos para inibição de genes de imunoglobulinas endógenas e produção de anticorpos idiótipos humanos transgénicos |
| US7887801B2 (en) | 2007-07-13 | 2011-02-15 | Topotarget Germany Ag | Optimized DNA and protein sequence of an antibody to improve quality and yield of bacterially expressed antibody fusion proteins |
| KR20100097691A (ko) * | 2007-11-12 | 2010-09-03 | 테라클론 사이언시스, 아이엔씨. | 인플루엔자의 치료 및 진단을 위한 조성물 및 방법 |
| AU2008328370A1 (en) * | 2007-11-22 | 2009-05-28 | Symphogen A/S | A method for characterization of a recombinant polyclonal protein |
| EP2088432A1 (en) | 2008-02-11 | 2009-08-12 | MorphoSys AG | Methods for identification of an antibody or a target |
| WO2010002911A2 (en) | 2008-06-30 | 2010-01-07 | H. Lee Moffitt Cancer Center And Research Institute, Inc. | Methods and materials for monitoring myeloma using quantitative mass spetrometry |
| NZ592308A (en) | 2008-09-30 | 2012-11-30 | Ablexis Llc | Non-human mammals for the production of chimeric antibodies |
| MX2011007660A (es) | 2008-12-18 | 2011-08-17 | Kingdon Craig R | Animales transgenicos no humanos que expresan anticuerpos humanizados y usos de los mismos. |
| ES2726702T3 (es) | 2009-01-15 | 2019-10-08 | Adaptive Biotechnologies Corp | Perfilado de la inmunidad adaptativa y métodos para la generación de anticuerpos monoclonales |
| TR201818431T4 (tr) | 2009-02-25 | 2019-01-21 | Ucb Biopharma Sprl | Antikorlar Üretmek İçin Usul |
| PL3241435T3 (pl) | 2009-07-08 | 2021-12-13 | Kymab Limited | Modele zwierzęce i cząsteczki terapeutyczne |
| US20110045534A1 (en) | 2009-08-20 | 2011-02-24 | Cell Signaling Technology, Inc. | Nucleic Acid Cassette For Producing Recombinant Antibodies |
| US8293483B2 (en) | 2009-09-11 | 2012-10-23 | Epitomics, Inc. | Method for identifying lineage-related antibodies |
| EP3251503B1 (en) | 2010-03-31 | 2024-03-20 | Ablexis, LLC | Genetic engineering of mice for the production of chimeric antibodies |
| EP2566984B1 (en) | 2010-05-07 | 2019-04-03 | The Board of Trustees of the Leland Stanford Junior University | Measurement and comparison of immune diversity by high-throughput sequencing |
| AU2011256290B2 (en) * | 2010-05-17 | 2014-06-12 | The Board Of Regents Of The University Of Texas System | Rapid isolation of monoclonal antibodies from animals |
| CN103228130B (zh) | 2010-06-17 | 2016-03-16 | 科马布有限公司 | 动物模型及治疗分子 |
-
2012
- 2012-03-09 KR KR1020197026655A patent/KR20190107761A/ko not_active Application Discontinuation
- 2012-03-09 CN CN201810154961.6A patent/CN108220297A/zh active Pending
- 2012-03-09 KR KR1020197000660A patent/KR20190006083A/ko not_active Application Discontinuation
- 2012-03-09 EP EP12711501.2A patent/EP2683736B1/en active Active
- 2012-03-09 EP EP17196465.3A patent/EP3345922A1/en not_active Withdrawn
- 2012-03-09 US US13/416,582 patent/US9920110B2/en active Active
- 2012-03-09 KR KR1020137026647A patent/KR101938343B1/ko active IP Right Grant
- 2012-03-09 WO PCT/US2012/028501 patent/WO2012122484A1/en unknown
- 2012-03-09 CN CN201280021326.1A patent/CN103842379A/zh active Pending
- 2012-03-09 CA CA2829606A patent/CA2829606C/en active Active
- 2012-03-09 AU AU2012225310A patent/AU2012225310B2/en active Active
- 2012-03-09 JP JP2013557906A patent/JP6211931B2/ja active Active
- 2012-03-09 ES ES12711501.2T patent/ES2666301T3/es active Active
- 2012-03-09 DK DK12711501.2T patent/DK2683736T3/en active
- 2012-03-09 HU HUE12711501A patent/HUE037095T2/hu unknown
-
2017
- 2017-09-13 JP JP2017175320A patent/JP6557709B2/ja not_active Expired - Fee Related
- 2017-10-12 AU AU2017245395A patent/AU2017245395B2/en active Active
-
2018
- 2018-01-19 US US15/875,615 patent/US20190002532A1/en not_active Abandoned
- 2018-11-30 HK HK18115369.1A patent/HK1256304A1/zh unknown
-
2019
- 2019-07-11 JP JP2019128863A patent/JP2019195335A/ja active Pending
- 2019-08-14 AU AU2019216660A patent/AU2019216660A1/en not_active Abandoned
Patent Citations (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN101679513A (zh) * | 2007-03-06 | 2010-03-24 | 西福根有限公司 | 用于治疗呼吸道合胞病毒感染的重组抗体 |
Non-Patent Citations (2)
| Title |
|---|
| BIRGIT OBERMEIER ET AL: "Matching of oligoclonal immunoglobulin transcriptomes ans proteomes of cerebrospinal fluid in multiple sclerosis", 《NATURE MEDICINE》, vol. 14, no. 6, 1 June 2008 (2008-06-01), pages 688 - 693, XP055026373, DOI: 10.1038/nm1714 * |
| SAI T REDDY ET AL: "Monoclonal antibodies isolated without screening by analyzing the variable-gene repertoire of plasma cells", 《NATURE BIOTECHNOLOGY》, vol. 28, no. 9, 30 September 2010 (2010-09-30), pages 965 - 969 * |
Cited By (15)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN105277638A (zh) * | 2014-07-22 | 2016-01-27 | 沈鹤霄 | 一种单克隆抗体IgG质谱测序方法 |
| CN104560979A (zh) * | 2015-01-20 | 2015-04-29 | 中国人民解放军第三军医大学 | 基于高通量测序构建鼠bcr重链文库的多重pcr引物和方法 |
| CN104560980A (zh) * | 2015-01-20 | 2015-04-29 | 中国人民解放军第三军医大学 | 基于高通量测序构建鼠BCR轻链Lamda文库的多重PCR引物和方法 |
| CN108348167A (zh) * | 2015-09-09 | 2018-07-31 | 优比欧迈公司 | 用于脑-颅面健康相关病症的源自微生物群系的诊断及治疗方法和系统 |
| CN106047857A (zh) * | 2016-06-01 | 2016-10-26 | 苏州金唯智生物科技有限公司 | 一种发掘特异性功能抗体的方法 |
| CN106047857B (zh) * | 2016-06-01 | 2020-04-03 | 苏州金唯智生物科技有限公司 | 一种发掘特异性功能抗体的方法 |
| CN107525934A (zh) * | 2016-06-21 | 2017-12-29 | 张效龙 | 一种测定血液中抗体氨基酸序列的方法 |
| CN110382696A (zh) * | 2017-03-07 | 2019-10-25 | 豪夫迈·罗氏有限公司 | 发现替代性抗原特异性抗体变体的方法 |
| CN110382696B (zh) * | 2017-03-07 | 2024-04-19 | 豪夫迈·罗氏有限公司 | 发现替代性抗原特异性抗体变体的方法 |
| CN111201238B (zh) * | 2017-07-21 | 2023-09-08 | 诺夫免疫股份有限公司 | 产生多特异性抗体混合物及其使用方法 |
| CN111201238A (zh) * | 2017-07-21 | 2020-05-26 | 诺夫免疫股份有限公司 | 产生多特异性抗体混合物及其使用方法 |
| CN109425662A (zh) * | 2017-08-23 | 2019-03-05 | 深圳华大基因研究院 | 一种鉴定蛋白的方法及系统 |
| CN108530533A (zh) * | 2018-04-13 | 2018-09-14 | 吉林大学 | 猪瘟病毒单克隆抗体hk24及医用用途 |
| CN110452296A (zh) * | 2019-07-16 | 2019-11-15 | 福州精锐生物技术有限公司 | 一种兔单克隆抗体制备方法 |
| WO2023184734A1 (zh) * | 2022-03-31 | 2023-10-05 | 台州恩泽医疗中心(集团) | 一种抗hla-g1,-g2,-g5及hla-g6异构体分子的单克隆抗体及其用途 |
Also Published As
| Publication number | Publication date |
|---|---|
| KR20140010436A (ko) | 2014-01-24 |
| JP6211931B2 (ja) | 2017-10-11 |
| KR101938343B1 (ko) | 2019-01-14 |
| JP2014509853A (ja) | 2014-04-24 |
| AU2017245395B2 (en) | 2019-05-16 |
| AU2017245395A2 (en) | 2017-11-23 |
| DK2683736T3 (en) | 2018-04-23 |
| CN108220297A (zh) | 2018-06-29 |
| AU2017245395A1 (en) | 2017-11-02 |
| EP2683736B1 (en) | 2018-01-17 |
| KR20190006083A (ko) | 2019-01-16 |
| JP2018027090A (ja) | 2018-02-22 |
| JP2019195335A (ja) | 2019-11-14 |
| US20190002532A1 (en) | 2019-01-03 |
| HK1256304A1 (zh) | 2019-09-20 |
| WO2012122484A1 (en) | 2012-09-13 |
| AU2019216660A1 (en) | 2019-09-05 |
| EP2683736A1 (en) | 2014-01-15 |
| CA2829606C (en) | 2020-06-02 |
| US20120308555A1 (en) | 2012-12-06 |
| KR20190107761A (ko) | 2019-09-20 |
| ES2666301T3 (es) | 2018-05-03 |
| JP6557709B2 (ja) | 2019-08-07 |
| CA2829606A1 (en) | 2012-09-13 |
| AU2012225310B2 (en) | 2017-08-03 |
| AU2012225310A1 (en) | 2013-09-26 |
| HUE037095T2 (hu) | 2018-08-28 |
| US9920110B2 (en) | 2018-03-20 |
| EP3345922A1 (en) | 2018-07-11 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| CN103842379A (zh) | 用于生成单克隆抗体的方法和试剂 | |
| CN107108746B (zh) | 能够中和具有替换凝固因子viii(fviii)的功能的活性的物质的抗体 | |
| CN103827145A (zh) | 相对于亲和性复合物的抗体 | |
| CN113512112A (zh) | 针对细胞内抗原的单结构域抗体 | |
| CN115485392A (zh) | 用于鉴别配体阻断性抗体及用于确定抗体效价的方法 | |
| AU2020331963A1 (en) | Anti-CD19 antibodies and uses thereof | |
| EP4017869A1 (en) | Anti-cd22 antibodies and uses thereof | |
| CN111201239A (zh) | 用于开发特异于表位翻译后修饰状态的抗体的方法和组合物 | |
| EP3072976B1 (en) | Method for determining and system for determining polypeptide bonding to target molecule | |
| US20220185911A1 (en) | Therapeutic antibodies for treating lung cancer | |
| CN117015302A (zh) | 抗原特异性抗体的鉴定和产生 | |
| TW202229328A (zh) | 抗原特異性抗體之鑑定及產生 | |
| US9518131B2 (en) | Generating metal ion binding proteins | |
| Georgiou et al. | Rapid isolation of monoclonal antibodies from animals | |
| US20110077164A1 (en) | Expression profiling platform technology | |
| Kuhlmann | Immunization, immune sera and the production of antibodies | |
| Saragozza | High-throughput antibody validation platforms |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| C06 | Publication | ||
| PB01 | Publication | ||
| C10 | Entry into substantive examination | ||
| SE01 | Entry into force of request for substantive examination | ||
| RJ01 | Rejection of invention patent application after publication |
Application publication date: 20140604 |
|
| RJ01 | Rejection of invention patent application after publication |