CN102076846A - 具有改善的底物特异性的pqq-依赖性葡萄糖脱氢酶的新变体 - Google Patents
具有改善的底物特异性的pqq-依赖性葡萄糖脱氢酶的新变体 Download PDFInfo
- Publication number
- CN102076846A CN102076846A CN200980124063.5A CN200980124063A CN102076846A CN 102076846 A CN102076846 A CN 102076846A CN 200980124063 A CN200980124063 A CN 200980124063A CN 102076846 A CN102076846 A CN 102076846A
- Authority
- CN
- China
- Prior art keywords
- spqqgdh
- glucose
- amino acid
- sugar
- site
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Pending
Links
- 239000000758 substrate Substances 0.000 title claims abstract description 38
- 230000001419 dependent effect Effects 0.000 title claims abstract description 6
- 108010050375 Glucose 1-Dehydrogenase Proteins 0.000 title claims description 7
- 238000000034 method Methods 0.000 claims abstract description 19
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 claims description 92
- 239000008103 glucose Substances 0.000 claims description 92
- 150000001413 amino acids Chemical class 0.000 claims description 44
- 235000000346 sugar Nutrition 0.000 claims description 42
- 241000894006 Bacteria Species 0.000 claims description 32
- 230000000694 effects Effects 0.000 claims description 32
- 108091034117 Oligonucleotide Proteins 0.000 claims description 27
- 238000003780 insertion Methods 0.000 claims description 24
- 230000037431 insertion Effects 0.000 claims description 24
- 108090000623 proteins and genes Proteins 0.000 claims description 23
- MMXZSJMASHPLLR-UHFFFAOYSA-N pyrroloquinoline quinone Chemical compound C12=C(C(O)=O)C=C(C(O)=O)N=C2C(=O)C(=O)C2=C1NC(C(=O)O)=C2 MMXZSJMASHPLLR-UHFFFAOYSA-N 0.000 claims description 19
- 230000001580 bacterial effect Effects 0.000 claims description 14
- 238000012216 screening Methods 0.000 claims description 14
- 241000588624 Acinetobacter calcoaceticus Species 0.000 claims description 8
- 230000006872 improvement Effects 0.000 claims description 8
- 241000589291 Acinetobacter Species 0.000 claims description 6
- 102000004169 proteins and genes Human genes 0.000 claims description 5
- 102000000584 Calmodulin Human genes 0.000 claims description 4
- 108010041952 Calmodulin Proteins 0.000 claims description 4
- 229910052739 hydrogen Inorganic materials 0.000 claims description 4
- 238000002703 mutagenesis Methods 0.000 claims description 4
- 231100000350 mutagenesis Toxicity 0.000 claims description 4
- 239000013598 vector Substances 0.000 claims description 3
- 238000004519 manufacturing process Methods 0.000 abstract description 12
- 108040005045 glucose dehydrogenase activity proteins Proteins 0.000 abstract 1
- WQZGKKKJIJFFOK-VFUOTHLCSA-N beta-D-glucose Chemical compound OC[C@H]1O[C@@H](O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-VFUOTHLCSA-N 0.000 description 89
- OWEGMIWEEQEYGQ-UHFFFAOYSA-N 100676-05-9 Natural products OC1C(O)C(O)C(CO)OC1OCC1C(O)C(O)C(O)C(OC2C(OC(O)C(O)C2O)CO)O1 OWEGMIWEEQEYGQ-UHFFFAOYSA-N 0.000 description 42
- 102000004190 Enzymes Human genes 0.000 description 42
- 108090000790 Enzymes Proteins 0.000 description 42
- GUBGYTABKSRVRQ-PICCSMPSSA-N Maltose Natural products O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)O[C@@H]1O[C@@H]1[C@@H](CO)OC(O)[C@H](O)[C@H]1O GUBGYTABKSRVRQ-PICCSMPSSA-N 0.000 description 42
- 229940088598 enzyme Drugs 0.000 description 42
- 235000001014 amino acid Nutrition 0.000 description 40
- 108020004414 DNA Proteins 0.000 description 32
- 238000012360 testing method Methods 0.000 description 28
- 239000000203 mixture Substances 0.000 description 19
- 239000012634 fragment Substances 0.000 description 17
- 238000006243 chemical reaction Methods 0.000 description 16
- -1 Hexose phosphate Chemical class 0.000 description 13
- 229910019142 PO4 Inorganic materials 0.000 description 13
- 239000010452 phosphate Substances 0.000 description 13
- 101710088194 Dehydrogenase Proteins 0.000 description 12
- 101710122864 Major tegument protein Proteins 0.000 description 12
- 101710148592 PTS system fructose-like EIIA component Proteins 0.000 description 12
- 101710169713 PTS system fructose-specific EIIA component Proteins 0.000 description 12
- 101710199973 Tail tube protein Proteins 0.000 description 12
- 238000005259 measurement Methods 0.000 description 12
- 108091008146 restriction endonucleases Proteins 0.000 description 9
- 239000000243 solution Substances 0.000 description 9
- GUBGYTABKSRVRQ-QUYVBRFLSA-N beta-maltose Chemical compound OC[C@H]1O[C@H](O[C@H]2[C@H](O)[C@@H](O)[C@H](O)O[C@@H]2CO)[C@H](O)[C@@H](O)[C@@H]1O GUBGYTABKSRVRQ-QUYVBRFLSA-N 0.000 description 8
- 230000008859 change Effects 0.000 description 8
- 238000013016 damping Methods 0.000 description 8
- 239000012530 fluid Substances 0.000 description 8
- 239000002773 nucleotide Substances 0.000 description 8
- 125000003729 nucleotide group Chemical group 0.000 description 8
- 239000000047 product Substances 0.000 description 8
- 125000003275 alpha amino acid group Chemical group 0.000 description 7
- 108020004705 Codon Proteins 0.000 description 6
- SRBFZHDQGSBBOR-LECHCGJUSA-N alpha-D-xylose Chemical compound O[C@@H]1CO[C@H](O)[C@H](O)[C@H]1O SRBFZHDQGSBBOR-LECHCGJUSA-N 0.000 description 6
- 210000004027 cell Anatomy 0.000 description 6
- 239000013612 plasmid Substances 0.000 description 6
- BASFCYQUMIYNBI-UHFFFAOYSA-N platinum Chemical compound [Pt] BASFCYQUMIYNBI-UHFFFAOYSA-N 0.000 description 6
- 238000000746 purification Methods 0.000 description 6
- 239000000126 substance Substances 0.000 description 6
- 230000009466 transformation Effects 0.000 description 6
- 229960003487 xylose Drugs 0.000 description 6
- 108091093088 Amplicon Proteins 0.000 description 5
- 241000620209 Escherichia coli DH5[alpha] Species 0.000 description 5
- 238000010790 dilution Methods 0.000 description 5
- 239000012895 dilution Substances 0.000 description 5
- 230000012010 growth Effects 0.000 description 5
- 230000001939 inductive effect Effects 0.000 description 5
- 108090000765 processed proteins & peptides Proteins 0.000 description 5
- 230000009257 reactivity Effects 0.000 description 5
- 230000007306 turnover Effects 0.000 description 5
- 102000004594 DNA Polymerase I Human genes 0.000 description 4
- 108010017826 DNA Polymerase I Proteins 0.000 description 4
- 241000588724 Escherichia coli Species 0.000 description 4
- GUBGYTABKSRVRQ-QKKXKWKRSA-N Lactose Natural products OC[C@H]1O[C@@H](O[C@H]2[C@H](O)[C@@H](O)C(O)O[C@@H]2CO)[C@H](O)[C@@H](O)[C@H]1O GUBGYTABKSRVRQ-QKKXKWKRSA-N 0.000 description 4
- 238000012408 PCR amplification Methods 0.000 description 4
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 4
- 230000003321 amplification Effects 0.000 description 4
- 239000008280 blood Substances 0.000 description 4
- 210000004369 blood Anatomy 0.000 description 4
- 229910052799 carbon Inorganic materials 0.000 description 4
- 239000013613 expression plasmid Substances 0.000 description 4
- 239000008101 lactose Substances 0.000 description 4
- JPXMTWWFLBLUCD-UHFFFAOYSA-N nitro blue tetrazolium(2+) Chemical compound COC1=CC(C=2C=C(OC)C(=CC=2)[N+]=2N(N=C(N=2)C=2C=CC=CC=2)C=2C=CC(=CC=2)[N+]([O-])=O)=CC=C1[N+]1=NC(C=2C=CC=CC=2)=NN1C1=CC=C([N+]([O-])=O)C=C1 JPXMTWWFLBLUCD-UHFFFAOYSA-N 0.000 description 4
- 235000018102 proteins Nutrition 0.000 description 4
- DVLFYONBTKHTER-UHFFFAOYSA-N 3-(N-morpholino)propanesulfonic acid Chemical compound OS(=O)(=O)CCCN1CCOCC1 DVLFYONBTKHTER-UHFFFAOYSA-N 0.000 description 3
- GUBGYTABKSRVRQ-CUHNMECISA-N D-Cellobiose Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)O[C@H]1O[C@@H]1[C@@H](CO)OC(O)[C@H](O)[C@H]1O GUBGYTABKSRVRQ-CUHNMECISA-N 0.000 description 3
- FBWADIKARMIWNM-UHFFFAOYSA-N N-3,5-dichloro-4-hydroxyphenyl-1,4-benzoquinone imine Chemical compound C1=C(Cl)C(O)=C(Cl)C=C1N=C1C=CC(=O)C=C1 FBWADIKARMIWNM-UHFFFAOYSA-N 0.000 description 3
- 108091028043 Nucleic acid sequence Proteins 0.000 description 3
- WQZGKKKJIJFFOK-PQMKYFCFSA-N alpha-D-mannose Chemical compound OC[C@H]1O[C@H](O)[C@@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-PQMKYFCFSA-N 0.000 description 3
- 230000015572 biosynthetic process Effects 0.000 description 3
- 239000003153 chemical reaction reagent Substances 0.000 description 3
- 238000002474 experimental method Methods 0.000 description 3
- 244000005700 microbiome Species 0.000 description 3
- 238000003199 nucleic acid amplification method Methods 0.000 description 3
- 229910052697 platinum Inorganic materials 0.000 description 3
- OFVLGDICTFRJMM-WESIUVDSSA-N tetracycline Chemical compound C1=CC=C2[C@](O)(C)[C@H]3C[C@H]4[C@H](N(C)C)C(O)=C(C(N)=O)C(=O)[C@@]4(O)C(O)=C3C(=O)C2=C1O OFVLGDICTFRJMM-WESIUVDSSA-N 0.000 description 3
- 229930101283 tetracycline Natural products 0.000 description 3
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Chemical compound O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 3
- IHPYMWDTONKSCO-UHFFFAOYSA-N 2,2'-piperazine-1,4-diylbisethanesulfonic acid Chemical compound OS(=O)(=O)CCN1CCN(CCS(O)(=O)=O)CC1 IHPYMWDTONKSCO-UHFFFAOYSA-N 0.000 description 2
- QKNYBSVHEMOAJP-UHFFFAOYSA-N 2-amino-2-(hydroxymethyl)propane-1,3-diol;hydron;chloride Chemical compound Cl.OCC(N)(CO)CO QKNYBSVHEMOAJP-UHFFFAOYSA-N 0.000 description 2
- 241000588626 Acinetobacter baumannii Species 0.000 description 2
- 229920001817 Agar Polymers 0.000 description 2
- GUBGYTABKSRVRQ-XLOQQCSPSA-N Alpha-Lactose Chemical compound O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@H]1O[C@@H]1[C@@H](CO)O[C@H](O)[C@H](O)[C@H]1O GUBGYTABKSRVRQ-XLOQQCSPSA-N 0.000 description 2
- 108091003079 Bovine Serum Albumin Proteins 0.000 description 2
- OKTJSMMVPCPJKN-UHFFFAOYSA-N Carbon Chemical compound [C] OKTJSMMVPCPJKN-UHFFFAOYSA-N 0.000 description 2
- CURLTUGMZLYLDI-UHFFFAOYSA-N Carbon dioxide Chemical compound O=C=O CURLTUGMZLYLDI-UHFFFAOYSA-N 0.000 description 2
- 102000016928 DNA-directed DNA polymerase Human genes 0.000 description 2
- 108010014303 DNA-directed DNA polymerase Proteins 0.000 description 2
- SXRSQZLOMIGNAQ-UHFFFAOYSA-N Glutaraldehyde Chemical compound O=CCCCC=O SXRSQZLOMIGNAQ-UHFFFAOYSA-N 0.000 description 2
- 239000007990 PIPES buffer Substances 0.000 description 2
- 229920002684 Sepharose Polymers 0.000 description 2
- 229910021607 Silver chloride Inorganic materials 0.000 description 2
- 108020005038 Terminator Codon Proteins 0.000 description 2
- 239000008272 agar Substances 0.000 description 2
- 125000000539 amino acid group Chemical group 0.000 description 2
- AVKUERGKIZMTKX-NJBDSQKTSA-N ampicillin Chemical compound C1([C@@H](N)C(=O)N[C@H]2[C@H]3SC([C@@H](N3C2=O)C(O)=O)(C)C)=CC=CC=C1 AVKUERGKIZMTKX-NJBDSQKTSA-N 0.000 description 2
- 229960000723 ampicillin Drugs 0.000 description 2
- 229960003311 ampicillin trihydrate Drugs 0.000 description 2
- 238000003556 assay Methods 0.000 description 2
- 229940098773 bovine serum albumin Drugs 0.000 description 2
- 239000003795 chemical substances by application Substances 0.000 description 2
- SUYVUBYJARFZHO-RRKCRQDMSA-N dATP Chemical compound C1=NC=2C(N)=NC=NC=2N1[C@H]1C[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 SUYVUBYJARFZHO-RRKCRQDMSA-N 0.000 description 2
- SUYVUBYJARFZHO-UHFFFAOYSA-N dATP Natural products C1=NC=2C(N)=NC=NC=2N1C1CC(O)C(COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 SUYVUBYJARFZHO-UHFFFAOYSA-N 0.000 description 2
- RGWHQCVHVJXOKC-SHYZEUOFSA-J dCTP(4-) Chemical compound O=C1N=C(N)C=CN1[C@@H]1O[C@H](COP([O-])(=O)OP([O-])(=O)OP([O-])([O-])=O)[C@@H](O)C1 RGWHQCVHVJXOKC-SHYZEUOFSA-J 0.000 description 2
- HAAZLUGHYHWQIW-KVQBGUIXSA-N dGTP Chemical compound C1=NC=2C(=O)NC(N)=NC=2N1[C@H]1C[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 HAAZLUGHYHWQIW-KVQBGUIXSA-N 0.000 description 2
- NHVNXKFIZYSCEB-XLPZGREQSA-N dTTP Chemical compound O=C1NC(=O)C(C)=CN1[C@@H]1O[C@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)[C@@H](O)C1 NHVNXKFIZYSCEB-XLPZGREQSA-N 0.000 description 2
- 238000000354 decomposition reaction Methods 0.000 description 2
- 230000030609 dephosphorylation Effects 0.000 description 2
- 238000006209 dephosphorylation reaction Methods 0.000 description 2
- 206010012601 diabetes mellitus Diseases 0.000 description 2
- VHJLVAABSRFDPM-QWWZWVQMSA-N dithiothreitol Chemical compound SC[C@@H](O)[C@H](O)CS VHJLVAABSRFDPM-QWWZWVQMSA-N 0.000 description 2
- 238000005516 engineering process Methods 0.000 description 2
- 238000006911 enzymatic reaction Methods 0.000 description 2
- 238000013467 fragmentation Methods 0.000 description 2
- 238000006062 fragmentation reaction Methods 0.000 description 2
- 238000013101 initial test Methods 0.000 description 2
- 230000000977 initiatory effect Effects 0.000 description 2
- 239000007788 liquid Substances 0.000 description 2
- 239000002609 medium Substances 0.000 description 2
- 238000002156 mixing Methods 0.000 description 2
- 238000010369 molecular cloning Methods 0.000 description 2
- 229950006238 nadide Drugs 0.000 description 2
- 230000003287 optical effect Effects 0.000 description 2
- 230000003647 oxidation Effects 0.000 description 2
- 238000007254 oxidation reaction Methods 0.000 description 2
- 238000005375 photometry Methods 0.000 description 2
- 238000002360 preparation method Methods 0.000 description 2
- 238000011160 research Methods 0.000 description 2
- 238000007789 sealing Methods 0.000 description 2
- 238000012163 sequencing technique Methods 0.000 description 2
- HKZLPVFGJNLROG-UHFFFAOYSA-M silver monochloride Chemical compound [Cl-].[Ag+] HKZLPVFGJNLROG-UHFFFAOYSA-M 0.000 description 2
- 239000011780 sodium chloride Substances 0.000 description 2
- 150000008163 sugars Chemical class 0.000 description 2
- PMUNIMVZCACZBB-UHFFFAOYSA-N 2-hydroxyethylazanium;chloride Chemical class Cl.NCCO PMUNIMVZCACZBB-UHFFFAOYSA-N 0.000 description 1
- RXGJTUSBYWCRBK-UHFFFAOYSA-M 5-methylphenazinium methyl sulfate Chemical compound COS([O-])(=O)=O.C1=CC=C2[N+](C)=C(C=CC=C3)C3=NC2=C1 RXGJTUSBYWCRBK-UHFFFAOYSA-M 0.000 description 1
- 229920000936 Agarose Polymers 0.000 description 1
- KYDYGANDJHFBCW-DRZSPHRISA-N Ala-Phe-Gln Chemical compound C[C@@H](C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CCC(=O)N)C(=O)O)N KYDYGANDJHFBCW-DRZSPHRISA-N 0.000 description 1
- IHMCQESUJVZTKW-UBHSHLNASA-N Ala-Phe-Val Chemical compound CC(C)[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)[C@H](C)N)CC1=CC=CC=C1 IHMCQESUJVZTKW-UBHSHLNASA-N 0.000 description 1
- YXXPVUOMPSZURS-ZLIFDBKOSA-N Ala-Trp-Leu Chemical compound C1=CC=C2C(C[C@@H](C(=O)N[C@@H](CC(C)C)C(O)=O)NC(=O)[C@H](C)N)=CNC2=C1 YXXPVUOMPSZURS-ZLIFDBKOSA-N 0.000 description 1
- BHFOJPDOQPWJRN-XDTLVQLUSA-N Ala-Tyr-Gln Chemical compound C[C@H](N)C(=O)N[C@@H](Cc1ccc(O)cc1)C(=O)N[C@@H](CCC(N)=O)C(O)=O BHFOJPDOQPWJRN-XDTLVQLUSA-N 0.000 description 1
- YEBZNKPPOHFZJM-BPNCWPANSA-N Ala-Tyr-Val Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](C(C)C)C(O)=O YEBZNKPPOHFZJM-BPNCWPANSA-N 0.000 description 1
- 102000002260 Alkaline Phosphatase Human genes 0.000 description 1
- 108020004774 Alkaline Phosphatase Proteins 0.000 description 1
- JSLGXODUIAFWCF-WDSKDSINSA-N Arg-Asn Chemical compound NC(N)=NCCC[C@H](N)C(=O)N[C@@H](CC(N)=O)C(O)=O JSLGXODUIAFWCF-WDSKDSINSA-N 0.000 description 1
- 108091033380 Coding strand Proteins 0.000 description 1
- 102000053602 DNA Human genes 0.000 description 1
- 108020005199 Dehydrogenases Proteins 0.000 description 1
- 229920002245 Dextrose equivalent Polymers 0.000 description 1
- 108010015776 Glucose oxidase Proteins 0.000 description 1
- 239000004366 Glucose oxidase Substances 0.000 description 1
- KOYUSMBPJOVSOO-XEGUGMAKSA-N Gly-Tyr-Ile Chemical compound [H]NCC(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H]([C@@H](C)CC)C(O)=O KOYUSMBPJOVSOO-XEGUGMAKSA-N 0.000 description 1
- 108010025076 Holoenzymes Proteins 0.000 description 1
- UFHFLCQGNIYNRP-UHFFFAOYSA-N Hydrogen Chemical compound [H][H] UFHFLCQGNIYNRP-UHFFFAOYSA-N 0.000 description 1
- BAWFJGJZGIEFAR-NNYOXOHSSA-O NAD(+) Chemical compound NC(=O)C1=CC=C[N+]([C@H]2[C@@H]([C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OC[C@@H]3[C@H]([C@@H](O)[C@@H](O3)N3C4=NC=NC(N)=C4N=C3)O)O2)O)=C1 BAWFJGJZGIEFAR-NNYOXOHSSA-O 0.000 description 1
- 235000003140 Panax quinquefolius Nutrition 0.000 description 1
- 240000005373 Panax quinquefolius Species 0.000 description 1
- 241000218636 Thuja Species 0.000 description 1
- 229920004890 Triton X-100 Polymers 0.000 description 1
- 239000013504 Triton X-100 Substances 0.000 description 1
- XJLXINKUBYWONI-DQQFMEOOSA-N [[(2r,3r,4r,5r)-5-(6-aminopurin-9-yl)-3-hydroxy-4-phosphonooxyoxolan-2-yl]methoxy-hydroxyphosphoryl] [(2s,3r,4s,5s)-5-(3-carbamoylpyridin-1-ium-1-yl)-3,4-dihydroxyoxolan-2-yl]methyl phosphate Chemical compound NC(=O)C1=CC=C[N+]([C@@H]2[C@H]([C@@H](O)[C@H](COP([O-])(=O)OP(O)(=O)OC[C@@H]3[C@H]([C@@H](OP(O)(O)=O)[C@@H](O3)N3C4=NC=NC(N)=C4N=C3)O)O2)O)=C1 XJLXINKUBYWONI-DQQFMEOOSA-N 0.000 description 1
- 239000002253 acid Substances 0.000 description 1
- 230000003213 activating effect Effects 0.000 description 1
- 239000000654 additive Substances 0.000 description 1
- 230000000996 additive effect Effects 0.000 description 1
- 239000002671 adjuvant Substances 0.000 description 1
- 150000001412 amines Chemical class 0.000 description 1
- 238000004082 amperometric method Methods 0.000 description 1
- QVGXLLKOCUKJST-UHFFFAOYSA-N atomic oxygen Chemical compound [O] QVGXLLKOCUKJST-UHFFFAOYSA-N 0.000 description 1
- 230000008033 biological extinction Effects 0.000 description 1
- 229910002092 carbon dioxide Inorganic materials 0.000 description 1
- 239000001569 carbon dioxide Substances 0.000 description 1
- 239000000969 carrier Substances 0.000 description 1
- 230000015556 catabolic process Effects 0.000 description 1
- 230000003197 catalytic effect Effects 0.000 description 1
- 239000006285 cell suspension Substances 0.000 description 1
- 238000005660 chlorination reaction Methods 0.000 description 1
- 238000010367 cloning Methods 0.000 description 1
- 238000004440 column chromatography Methods 0.000 description 1
- 230000000295 complement effect Effects 0.000 description 1
- 150000001875 compounds Chemical class 0.000 description 1
- 229920001940 conductive polymer Polymers 0.000 description 1
- 238000001816 cooling Methods 0.000 description 1
- 238000012258 culturing Methods 0.000 description 1
- 238000004042 decolorization Methods 0.000 description 1
- 238000006731 degradation reaction Methods 0.000 description 1
- 238000012217 deletion Methods 0.000 description 1
- 230000037430 deletion Effects 0.000 description 1
- 238000004925 denaturation Methods 0.000 description 1
- 230000036425 denaturation Effects 0.000 description 1
- 238000013461 design Methods 0.000 description 1
- 238000001514 detection method Methods 0.000 description 1
- 238000011161 development Methods 0.000 description 1
- 238000003745 diagnosis Methods 0.000 description 1
- 238000002405 diagnostic procedure Methods 0.000 description 1
- 238000000502 dialysis Methods 0.000 description 1
- 230000029087 digestion Effects 0.000 description 1
- 150000002016 disaccharides Chemical class 0.000 description 1
- 238000012850 discrimination method Methods 0.000 description 1
- 208000016097 disease of metabolism Diseases 0.000 description 1
- 238000006073 displacement reaction Methods 0.000 description 1
- 238000004090 dissolution Methods 0.000 description 1
- 239000012154 double-distilled water Substances 0.000 description 1
- 239000003814 drug Substances 0.000 description 1
- 230000002255 enzymatic effect Effects 0.000 description 1
- 230000003203 everyday effect Effects 0.000 description 1
- 239000013604 expression vector Substances 0.000 description 1
- KTWOOEGAPBSYNW-UHFFFAOYSA-N ferrocene Chemical compound [Fe+2].C=1C=C[CH-]C=1.C=1C=C[CH-]C=1 KTWOOEGAPBSYNW-UHFFFAOYSA-N 0.000 description 1
- 239000000499 gel Substances 0.000 description 1
- 150000002303 glucose derivatives Chemical class 0.000 description 1
- 229940116332 glucose oxidase Drugs 0.000 description 1
- 235000019420 glucose oxidase Nutrition 0.000 description 1
- 125000002791 glucosyl group Chemical group C1([C@H](O)[C@@H](O)[C@H](O)[C@H](O1)CO)* 0.000 description 1
- 125000000404 glutamine group Chemical group N[C@@H](CCC(N)=O)C(=O)* 0.000 description 1
- PCHJSUWPFVWCPO-UHFFFAOYSA-N gold Chemical compound [Au] PCHJSUWPFVWCPO-UHFFFAOYSA-N 0.000 description 1
- 229910052737 gold Inorganic materials 0.000 description 1
- 239000010931 gold Substances 0.000 description 1
- 238000004442 gravimetric analysis Methods 0.000 description 1
- 239000001963 growth medium Substances 0.000 description 1
- 150000002402 hexoses Chemical group 0.000 description 1
- 239000001257 hydrogen Substances 0.000 description 1
- 210000003000 inclusion body Anatomy 0.000 description 1
- 239000003112 inhibitor Substances 0.000 description 1
- 238000011081 inoculation Methods 0.000 description 1
- 238000011835 investigation Methods 0.000 description 1
- 150000002596 lactones Chemical class 0.000 description 1
- 239000000463 material Substances 0.000 description 1
- 239000011159 matrix material Substances 0.000 description 1
- 239000012528 membrane Substances 0.000 description 1
- 208000030159 metabolic disease Diseases 0.000 description 1
- 230000000813 microbial effect Effects 0.000 description 1
- 231100000219 mutagenic Toxicity 0.000 description 1
- 230000003505 mutagenic effect Effects 0.000 description 1
- 229930027945 nicotinamide-adenine dinucleotide Natural products 0.000 description 1
- BOPGDPNILDQYTO-NNYOXOHSSA-N nicotinamide-adenine dinucleotide Chemical group C1=CCC(C(=O)N)=CN1[C@H]1[C@H](O)[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OC[C@@H]2[C@H]([C@@H](O)[C@@H](O2)N2C3=NC=NC(N)=C3N=C2)O)O1 BOPGDPNILDQYTO-NNYOXOHSSA-N 0.000 description 1
- 238000011017 operating method Methods 0.000 description 1
- 239000001301 oxygen Substances 0.000 description 1
- 229910052760 oxygen Inorganic materials 0.000 description 1
- 238000001050 pharmacotherapy Methods 0.000 description 1
- NBIIXXVUZAFLBC-UHFFFAOYSA-K phosphate Chemical compound [O-]P([O-])([O-])=O NBIIXXVUZAFLBC-UHFFFAOYSA-K 0.000 description 1
- 229920000642 polymer Polymers 0.000 description 1
- 229920001184 polypeptide Polymers 0.000 description 1
- 229920001296 polysiloxane Polymers 0.000 description 1
- 239000013641 positive control Substances 0.000 description 1
- 239000002244 precipitate Substances 0.000 description 1
- 230000008569 process Effects 0.000 description 1
- 102000004196 processed proteins & peptides Human genes 0.000 description 1
- 239000000700 radioactive tracer Substances 0.000 description 1
- 238000002708 random mutagenesis Methods 0.000 description 1
- 239000000376 reactant Substances 0.000 description 1
- 239000011541 reaction mixture Substances 0.000 description 1
- 230000009467 reduction Effects 0.000 description 1
- 150000003839 salts Chemical class 0.000 description 1
- 238000000926 separation method Methods 0.000 description 1
- 238000012882 sequential analysis Methods 0.000 description 1
- 238000004904 shortening Methods 0.000 description 1
- 239000006228 supernatant Substances 0.000 description 1
- 230000008093 supporting effect Effects 0.000 description 1
- 230000001225 therapeutic effect Effects 0.000 description 1
- 238000007669 thermal treatment Methods 0.000 description 1
- 230000001131 transforming effect Effects 0.000 description 1
- 238000013519 translation Methods 0.000 description 1
- BYGOPQKDHGXNCD-UHFFFAOYSA-N tripotassium;iron(3+);hexacyanide Chemical compound [K+].[K+].[K+].[Fe+3].N#[C-].N#[C-].N#[C-].N#[C-].N#[C-].N#[C-] BYGOPQKDHGXNCD-UHFFFAOYSA-N 0.000 description 1
- GPRLSGONYQIRFK-MNYXATJNSA-N triton Chemical compound [3H+] GPRLSGONYQIRFK-MNYXATJNSA-N 0.000 description 1
- 238000011144 upstream manufacturing Methods 0.000 description 1
- 210000002700 urine Anatomy 0.000 description 1
- 238000012800 visualization Methods 0.000 description 1
- 238000005406 washing Methods 0.000 description 1
- 239000002023 wood Substances 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/001—Enzyme electrodes
- C12Q1/005—Enzyme electrodes involving specific analytes or enzymes
- C12Q1/006—Enzyme electrodes involving specific analytes or enzymes for glucose
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/0004—Oxidoreductases (1.)
- C12N9/0006—Oxidoreductases (1.) acting on CH-OH groups as donors (1.1)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/26—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving oxidoreductase
- C12Q1/32—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving oxidoreductase involving dehydrogenase
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y101/00—Oxidoreductases acting on the CH-OH group of donors (1.1)
- C12Y101/01—Oxidoreductases acting on the CH-OH group of donors (1.1) with NAD+ or NADP+ as acceptor (1.1.1)
- C12Y101/01069—Gluconate 5-dehydrogenase (1.1.1.69)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y101/00—Oxidoreductases acting on the CH-OH group of donors (1.1)
- C12Y101/05—Oxidoreductases acting on the CH-OH group of donors (1.1) with a quinone or similar compound as acceptor (1.1.5)
- C12Y101/05002—Quinoprotein glucose dehydrogenase (1.1.5.2)
Landscapes
- Chemical & Material Sciences (AREA)
- Organic Chemistry (AREA)
- Life Sciences & Earth Sciences (AREA)
- Health & Medical Sciences (AREA)
- Engineering & Computer Science (AREA)
- Zoology (AREA)
- Wood Science & Technology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Genetics & Genomics (AREA)
- Biochemistry (AREA)
- General Health & Medical Sciences (AREA)
- General Engineering & Computer Science (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Molecular Biology (AREA)
- Microbiology (AREA)
- Biotechnology (AREA)
- Analytical Chemistry (AREA)
- Physics & Mathematics (AREA)
- Biophysics (AREA)
- Immunology (AREA)
- Biomedical Technology (AREA)
- Medicinal Chemistry (AREA)
- Emergency Medicine (AREA)
- Enzymes And Modification Thereof (AREA)
- Measuring Or Testing Involving Enzymes Or Micro-Organisms (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Apparatus Associated With Microorganisms And Enzymes (AREA)
Abstract
本发明涉及新的PQQ依赖性可溶性葡萄糖脱氢酶(sPQQGDH),与野生型相比较而言,其具有增加的底物特异性,而且还涉及用于其生产和鉴别的方法。
Description
技术领域
本发明的主题是PQQ-依赖性葡萄糖脱氢酶 (sPQQGDH),它们的生产和鉴别方法,编码根据本发明的sPQQGDH的基因,生产该基因的载体,以及包含根据本发明的葡萄糖脱氢酶的葡萄糖传感器。
背景技术
葡萄糖的酶测定在医学诊断中是非常重要的。这里,检测尿或血液中的葡萄糖以诊断或监测代谢性疾病的进展是必需的。在糖尿病(也称为糖尿病)的情况中,每天可能需要多次测定血糖浓度。这可以通过市场上绝大多数的计量器通过酶方法实现,在酶方法中葡萄糖被氧化,由此得到的氢(2H+和2e-)进行电流分析定量(P. Vadgama,J.Med.Eng Technol. 5 [6],293-298 (1981),K.S. Chua 和I.K. Tan,Clin.Chem. 24 [1],150-152 (1978))。
存在许多适合这种目的的各种酶,但是这些酶与各种辅助因子共同作用进行电子的主要接收,并且还具有不同的生化性质诸如特异性、稳定性或转换率。酶的转换率通常以活性单位(U)进行报道;在葡萄糖-氧化酶的情况中,比活性 (U/mg) 通常是指在确定的反应条件下每分钟被1毫克酶氧化的以微摩尔计的葡萄糖的量。
通常使用的酶为葡萄糖脱氢酶(GDH)。在NAD(P)+-依赖性GDH的情况中,辅助因子为烟酰胺腺嘌呤二核苷酸(NAD+)或烟酰胺腺嘌呤磷酸二核苷酸(NADP+) (H. E.
Pauly 和G. Pfleiderer,Hoppe Seylers. Z. Physiol
Chem. 356 [10],1613-1623 (1975)),二者均为相当不稳定的必须添加到反应中的化合物,因为它们不与酶结合。
另一类葡萄糖脱氢酶含有作为电子受体与酶结合的吡咯并喹啉醌 (PQQ)。已经描述了这种类型中两种结构相互不同的葡萄糖脱氢酶:膜锚定型,mPQQGDH (A.M Cleton-Jansen等,J. Bacteriol. 170 [5],2121-2125 (1988)),和较小的可溶型,sPQQGDH (A.M.
Cleton-Jansen等,Mol.Gen.Genet. 217 [2-3],430-436 (1989))。由于后者的高比活性、其对氧的不敏感性以及紧密地结合,其为稳定的辅助因子,为特别适合生产用于葡萄糖测定的测试系统的酶。因此在许多商品化的血糖计量器中,使用这种酶,尽管与提到的其他酶相比,这种酶显示更低的底物特异性。由于最后提到的限制,当然希望拥有具有更高底物特异性的这种酶的变体。
除了底物葡萄糖外,sPQQGDH还将其他糖转化为相应的内酯,优选二糖,其中还原糖为葡萄糖,但也有其他还原糖。由于这种糖正常不存在于人的血液中,基于sPQQGDH的酶测试通常得到对应于葡萄糖浓度的结果。然而,某些医学治疗包含施用物质,该物质的一种分解产物为麦芽糖,麦芽糖被sPQQGDH氧化并且因此不希望地影响测量结果(T.G. Schleis,Pharmacotherapy 27 [9],1313-1321 (2007))。在诊断方法中或者作为药物的佐剂施用木糖和半乳糖时,测定这些糖自身。因此以适当的方式抑制sPQQGDH的辅助活性是有意义的。
在文献以及在专利说明书中,描述了许多通过引入突变来改善sPQQGDH的底物特异性的尝试 (S. Igarashi等,Biomol. Eng 21 [2],81-89 (2004))。为此,在酶的催化位点(EP1666586A1)以及在其他位点(US7132270B2)的区域内的单个氨基酸通过定向诱变被其他氨基酸取代,或者通过插入来插入单个氨基酸(EP1367120A2)。此外,描述了各种突变的组合(EP1666586A1,US7132270B2)。另外,已经描述了在sPQQGDH的一个位点插入两个氨基酸的变体。然而,结果没有获得底物特异性的显著改善(WO2006085509)。
尽管存在这些结果,但仍然需要sPQQGDH的变体,因为至今还未描述过在与葡萄糖足够高的反应性下能够充分识别葡萄糖和其他糖的变体。这里重要的是,更特别地,即使在葡萄糖和其他糖的混合物的存在下,酶使葡萄糖浓度可靠地测定的能力。所描述的变体没有充分地显示这种性质。
发明内容
考虑到现有技术,因此本发明的目的是提供使可靠的葡萄糖测定成为可能的葡萄糖脱氢酶,即使在葡萄糖和其他糖的混合物的存在下。更特别地,本发明的目的是提供葡萄糖脱氢酶,使用该葡萄糖脱氢酶可以在麦芽糖的存在下可靠地测定葡萄糖。此外,本发明的目的是提供可以生产和鉴别这种葡萄糖脱氢酶的方法,在其他糖的存在下,更特别地在麦芽糖的存在下,该葡萄糖脱氢酶特别适合用于葡萄糖的测定。
现已惊奇地发现,在底物结合区域的范围内插入3-5个氨基酸的sPQQ 葡萄糖脱氢酶,与酶的原型相比显示改善的葡萄糖特异性。
因此,本发明的主题是与酶的原型相比具有改善的葡萄糖特异性的sPQQ 葡萄糖脱氢酶,其特征在于,在底物结合区域的范围内插入3-5个氨基酸。
可以通过所述的天然存在的野生型菌株通过诱变获得的基因编码根据本发明的sPQQGDH,诸如醋酸钙不动杆菌(Acinetobacter
calcoaceticus) LMD 79.41 (K. Kojima K.等,Biotechnology Letters 22,1343-1347 (2000)),以及不动杆菌(Acinetobacter)属的其他菌株(P.J. Bouvet P.J. 和O.M. Bouvet,Res. Microbiol. 140 [8],531-540 (1989)),更特别地具有增强的热稳定性的那些,诸如在EP1623025A1中描述的不动杆菌属菌株。优选的菌株为EP1623025A1中描述的不动杆菌属菌株PT16、KOZ62、KOZ65、PTN69、KG106、PTN26、PT15、KGN80、KG140、KGN34、KGN25和KGN100;更特别优选的是菌株KOZ65。
为了能够获得根据本发明的sPQQGDH,密码子必须插入到对应于适合这种方式的sPQQGDH基因中的底物结合位点的位置。许多sPQQGDH的空间结构是已知的并且储存于公共数据库中 (A. Oubrie等,J. Mol. Biol. 289 [2],319-333 (1999);A. Oubrie等,EMBO J. 18 [19],5187-5194 (1999))。因此,可以估算各个氨基酸位置与结合底物的距离。这种估算,例如,可能使用蛋白质的可视化程序,诸如由互联网上可以获得的软件程序Cn3D(http://www.ncbi.nlm.nih.gov/Structure/CN3D/cn3d.shtml)。公开的X-射线结构的序列是基于醋酸钙不动杆菌的sPQQGDH,其长度为455个氨基酸。更优选的来自菌株KOZ65的sPQQGDH的原型具有456个氨基酸,就像鲍氏不动杆菌(Acinetobacter baumannii)的酶一样。在两种情况中,这样的原因是由于位置289之后其他的谷氨酰胺残基。以下是与更短的、在X-射线结构中所用的sPQQGDH序列有关的各个氨基酸位置的细节,所述sPQQGDH来源于菌株醋酸钙不动杆菌LMD 79.41。据报道位于那里的氨基酸在该位置前面的氨基酸的单字母密码中。
距离底物不大于5 埃的氨基酸区域,诸如,例如,位置Q76、D143、Q168、Y343和各自邻近的氨基酸残基,对于诱变而言是优选的。因此,3个或更多个氨基酸的插入可以,例如,在一个特定的位置之前或之后出现,以便改变底物结合位点的空间结构。更优选的是在位置Q168和L169之间的其他氨基酸的插入。在该位点具有插入的KOZ65的变体主要令人惊奇地表现出,与葡萄糖相比大比例的显著降低的对麦芽糖的活性。特别优选的是在位置Q168和L169之间具有表1所列的插入序列的sPQQGDH。作为比较,野生型显示于第一行中。如表1所概述,根据本发明的sPQQGDH具有增加的底物特异性(更低的麦芽糖/葡萄糖比和葡萄糖/麦芽糖混合物中更低的葡萄糖干扰值)。
表 1:
本发明的主题还进一步是用于生产和鉴别 sPQQGDH的方法,所述sPQQGDH与野生型相比显示增强的底物特异性。通过定域随机诱变,根据本发明的方法可以生成多种变体,该变体在sPQQGDH 序列的所需位点具有3个、4个、5个或更多个插入的氨基酸。将生成的酶进行底物特异性筛选,选择最好的变体。
根据本发明的方法的特征在于,在第一步中生产载体,在该载体中在所需插入位点之前和之后的合适序列区域已经被删除并且被该载体的独特限制位点所替代。在第二步中向使用该限制性内切酶开放的载体-DNA 中插入双链寡核苷酸序列,以便得到基因产品,所述双链寡核苷酸序列通过PCR扩增生产并且除了载体中删除的序列外还含有所需的插入,所述基因产品除了在所需位点的3个或更多个氨基酸的插入外,对于其氨基酸序列,不含有其他改变。借助PCR由合成的寡核苷酸生产的待插入的双链片段含有3个或更多个用于改变底物特异性的其他随机密码子,以及载体中删除的密码子。为此所选择的密码子可以特别好地表达于E. coli.中。因此除了插入区域外,该核苷酸序列与KOZ65的sPQQGDH-基因的核苷酸序列不同,而不是与氨基酸序列不同。
使用3个其他氨基酸的插入时,8000个酶变体是可能的;在使用4个其他氨基酸时,数量为160,000;和在使用5个其他氨基酸时,存在320万个变体。因此除了完全随机的序列,诱变的寡核苷酸的合成可以利用,诸如在某些或所有位置含有对于每个待插入氨基酸的简并密码子的序列。可以这样使用以便减少所有可能性的数量,避免终止密码子或有利于特定位置的个别氨基酸。
在根据本发明方法的第三步中,将以如上所述方式生产的重组载体转换到合适的宿主细菌中,并且该宿主细菌在合适的生长培养基中增殖。由微生物克隆基因的方法和它们在另外的宿主中的表达以及分离和改变核苷酸的技术是本领域技术人员已知的(参见,例如,Molecular Cloning – A Laboratory Manual;第1卷; Sambrook,J.和Russel,D.W.;第3版,CSHL Press 2001)。培养微生物和从其中纯化重组体酶也是本领域已知的(参见同上;第3卷)。
在根据本发明方法的第四步中,测试菌落对各种糖的活性,由此鉴别出最好的。根据本发明,本文不但完成了对各种糖的活性的测试,而且还完成了各种糖的干扰的测试。
可以以比活性测定该活性,即,每时间单位通过确定量的酶转换的转换率。在葡萄糖氧化酶的情况中,比活性 (U/mg) 通常是指在确定的反应条件下每分钟被1毫克酶氧化的以微摩尔计的葡萄糖的量。相对于酶,葡萄糖通常是过量存在的,以便消除葡萄糖浓度对反应速率的影响。通过随时间示踪试剂(Edukts)的分解或产物的形成确定转换率。随时间示踪可以通过例如光度测定进行。为了确定反应速率和转换率, 可以参考物理化学教科书(例如,Peter Atkins,Physical Chemistry,W. H. Freeman & Co;第7版,2002)。
最初,分离各个菌落,使用葡萄糖作为底物进行酶试验 (确定比活性),使用第二种糖例如麦芽糖作为底物进行第二酶试验。在对葡萄糖具有最高活性而同时对第二种糖(例如,麦芽糖)具有最小活性的菌落的情况中,还测定了对葡萄糖和第二种糖(例如,麦芽糖)的混合物的活性。这是因为其特别出现在对作为唯一底物的麦芽糖具有非常低的相对反应性的某些变体的研究中,令人惊奇地,变体的这种性质并不总是伴有独立的性质,即当葡萄糖和麦芽糖两种糖作为混合物存在时对葡萄糖和麦芽糖的可靠识别。这会导致存在于该混合物中的葡萄糖浓度的高估和低估。因此在寻找用于测定测试样品中葡萄糖浓度的酶中,对葡萄糖和其他糖的混合物的高葡萄糖特异性是至关重要的,或者换句话说,所讨论的糖的尽可能低的干扰。糖的干扰通过由葡萄糖和干扰的糖的混合物测定的酶活性减去由相同浓度的葡萄糖单独测定的活性,标准化至该葡萄糖活性进行计算(参见等式1)。
(等式1)
在等式1中,I 为葡萄糖与其他糖的干扰,V参比为葡萄糖被所测定的酶分解的速率,V混合物为葡萄糖在与各种情况下的其他糖的混合物中的分解速率。该速率可以,例如通过光度测定确定(为此,参见实施例 3、4、7和9)。
在本文中正值表示对混合物的酶活性高于对葡萄糖单独的酶活性,即,两种糖明显都被氧化。负值可以解释为干扰糖也被氧化,但结果降低了酶对葡萄糖的活性的作用。两种作用对用于特异性定量葡萄糖的酶而言都是不希望的。
根据本发明的方法可以生产和鉴别sPQQGDH的变体,其中与酶的原型相比,改变了底物特异性。还可以修饰根据本发明的方法和/或将其扩展到并非底物特异性或除了底物特异性之外的作用,测试其他生化性质,诸如,例如,热稳定性,因此,鉴别与原型相比并且对于其他生化性质改善的变体。根据本发明的方法因此能够选择具有特别有利性质的sPQQGDH的变体。
本发明的主题还是编码根据本发明的sPQQGDH的基因,和用于生成该基因的载体。
本发明的主题还是用于测定葡萄糖的包含根据本发明的sPQQGDH的葡萄糖-传感器。基于葡萄糖脱氢酶的葡萄糖-传感器的设计和操作方式(Funktionsweise)是本领域技术人员由现有技术已知的(参见,例如,EP1146332 A1)。根据这些,这种葡萄糖-传感器包含电极系统,该电极系统在绝缘板上包括工作电极、对电极和参比电极,并且支承酶反应层,所述酶反应层含有葡萄糖脱氢酶和与电极系统接触的电子受体。
合适的工作电极包括碳、金、铂和类似的电极,根据本发明的酶通过交联剂固定在电极上:埋入聚合物基质中,使用透析膜包裹,使用光致交联聚合物、导电聚合物或氧化还原聚合物,将酶固定于聚合物中或者使用包括二茂络铁或其衍生物或其任意组合的电子传递体吸附到电极上。根据本发明的sPQQGDH优选以全酶形式固定在电极上,虽然它们也可以作为酶蛋白固定,PQQ作为分离层或在溶液中提供。根据本发明的sPQQGDH通常使用戊二醛固定在碳电极上,然后使用含有胺的试剂处理,以便封闭戊二醛。例如,铂电极,可以用作对电极,例如Ag/AgCl电极,可以用作参比电极。
可以如下测量葡萄糖的量:将PQQ、CaCl2和介质加入到含有缓冲液的恒温细胞中, 并置于恒温下。合适的介质包括,例如,铁氰化钾和吩嗪硫酸甲酯。根据本发明的sPQQGDH已经固定在其上的电极用作与对电极 (例如,铂电极)和参比电极 (例如,Ag/AgCl 电极)组合的工作电极。在应用恒定的电压在工作电极上建立稳定的电流后,加入含有葡萄糖的样品,以便测量电流强度的增加。可以由使用标准浓度的葡萄糖溶液建立的标准曲线读出样品的葡萄糖量。
下文借助实施例更详细地解释本发明,但本发明不限于实施例。
具体实施方式
实施例
下列实施例中引用的化学品是由例如Fa. Sigma-Aldrich或每种情况下指定的公司商业可获得的。
实施例1:用于在Q168和L169之间插入其他氨基酸的载体的生产
为了生产根据本发明的变体,必须在表达质粒中提供用于sPQQGDH的基因,该表达质粒可以在微生物中增殖以允许sPQQGDH的诱导表达,并且该表达质粒可以用于实施所需的突变。许多可以用于该目的的质粒和微生物是本领域技术人员已知的。在该实施例以及所有其他实施例中,使用基于商业可获得的载体pASK-IBA3 (IBA GmbH,Göttingen)的构建体。可以如下将菌株不动杆菌属 KOZ65的sPQQGDH基因克隆到pASK-IBA3中。根据生产商的说明,使用两种寡核苷酸 GDH-U3
(5'-TGGTAGGTCTCAAATGAATAAACATTTATTGGC TAAAATTAC-3';SEQ ID 19) 和GDH-L5 (5’-TTCAGCTCTGAGCTTTATATGTAAATCTAATC-3’;SEQ ID 20),借助PCR反应(Phusion DNA 聚合酶,Finnzymes Oy) 由不动杆菌属 KOZ65的基因组DNA生成1.46 kb长的DNA片段。提纯该片段并随后使用限制性酶BsaI培养。使用限制性内切酶HindIII开放载体pASK-IBA3。通过热处理使限制性内切酶变性,在dATP、dCTP、dGTP和dTTP的存在下使用T4 DNA多聚酶填充得到的DNA突出端。随后提纯DNA并使用限制性酶BsaI培养。清除这样释放的114 bp长的片段;将具有KOZ65的sPQQGDH基因的PCR 扩增子连接到3.1 kb长的载体片段中。将连接产物转化到E. coli菌株DH5α的感受态细胞中。以这种方式生产的载体 pAI3B-KOZ65适合KOZ65型的sPQQGDH的诱导性表达。载体 pAI3B-KOZ65的核苷酸序列报道于SEQ ID 21下;其编码的sPQQGDH的氨基酸序列报道于SEQ ID 22下。
为了能够在Q168和L169之间插入3个或更多个氨基酸,在初期生产载体 pAI3B-KOZ65-U。为此,载体pAI3B-KOZ65的质粒DNA用作使用引物IBA-For (5’-TAGAGTTATTTTACCACTCCCT-3’;SEQ ID 23) 和Eco109-Up (5’-GGTTAGGTCCCTGATCACCAATCG-3’;SEQ ID 24)的PCR扩增A的模板以及用作使用引物BfuA-US1 (5’-CACAGGTACACCTGCCGCCACT-3’;SEQ ID 25) 和Eco109-Down (5’-TACGAGGACCCAACAGGAACTGAGC-3’;SEQ ID 26)的PCR扩增B的模板。提纯由A得到的扩增子,使用限制性内切酶 XbaI和EcoO109I剪切。分离得到的588个碱基对(bp)长的片段。提纯由B得到的扩增子,使用限制性内切酶BfuAI和EcoO109I剪切。同样分离得到的353bp长的片段。使用限制性内切酶 BfuAI和XbaI剪切载体 pAI3B-KOZ65。在琼脂糖凝胶上分离得到的3567 bp长的片段。根据生产商的说明,对概述步骤使用来自Firmen New England Biolabs
GmbH (Frankfurt)、Invitrogen (Karlsruhe)、Qiagen (Hilden)和Roche Diagnostics (Mannheim)的试剂盒和试剂。由Sambrook和Russell的步骤文集(Molecular Cloning – A Laboratory Manual; Sambrook,J. 和Russel,D.W.;第3版,CSHL Press 2001)获得其他方法。
随后连接3个片段并通过化学转化将其转移到E. coli-DH5α中。由12个菌落制备质粒DNA,随后通过合适的限制性内切酶消化进行检查以确定是否得到所需的质粒,在9种分离物中是这样的。通过对这些克隆中的四种进行测序,证实在所有的情况下除了图1e概述的改变外没有发生其他改变。
载体 pAI3B-KOZ65-U (SEQ ID 27)仅含有一个唯一的EcoO109I 限制位点 (RG’GNCCY)。EcoO109I生成3 bp长的5’ 突出端,其中中间核苷酸是任意的;在pAI3B-KOZ65-U的情况中,其在编码链上是A。在pAI3B-KOZ65-U中删除编码氨基酸R166-P182的DNA以及其他核苷酸,导致阅读框中移位。因此尝试表达该基因得到没有功能的缩短的多肽(SEQ ID 28)。如实施例2中更详细地描述的那样,该载体适用于掺入合成的、双链DNA片段,该片段导致在位置Q168和L169之间具有插入的功能sPQQGDH基因的复位。
实施例2 – 生产sPQQGDH 插入突变体
为了生产在位置Q168和L169之间具有4个其他氨基酸的插入突变体, 使用4种合成寡核苷酸:EarV2-Random (5’-CGACGTAACCAGNNKNNKNNKNNKCTGGCTTACCTG-3’;SEQ ID 29)、EarV2-Lower (5’-GTGTGCTGTGCCTGGTTCGGCAGGAACAGGTAAGCCAG-3’;SEQ ID 30)、EarV2-UpAmp (5’-CCTACCTACGACTCTTCCGACGTAACCAG-3’;SEQ ID 31)和EarV2-LoAmp (5’-CCATGCTCTTCAGTCGGAGTGTGCTGTGCC-3’;SEQ ID 32)。在图2中,对由这些寡核苷酸生产双链插入片段的策略进行了图解说明。在这种情况下,4种所插入的氨基酸序列是随机的:NNK NNK NNK NNK。N在寡核苷酸的合成中在各自位置上是所有4种核苷酸的混合物(dATP (A)、dCTP (C)、dGTP (G)、dTTP (T)),而K为G或T。序列NNK使得所有20种氨基酸具有密码子成为可能,但仅针对三个终止密码子中的一个,降低不完全基因产物的可能性。在个别位置上的核苷酸的其他组合是自由选择的,为了例如限制密码子位置上的可能的氨基酸。寡核苷酸EarV2-Random 含有编码R166-L172的序列;代表互补链的寡核苷酸EarV2-Lower在L169-L172区域内重叠,并且一直延伸到T181。寡核苷酸EarV2-UpAmp在3’端与寡核苷酸EarV2-Random的5’端重叠12bp, 而寡核苷酸EarV2-LoAmp同样在3’端与寡核苷酸EarV2-Lower的5’端重叠12bp。最后提到的寡核苷酸各自含有EarI 限制位点 (CTCTTCN’NNN)。
首先,在PCR机器(GeneAmp 9600,Perkin-Elmer)中在10 mM Tris-HCl(25℃下pH 7.9)、10 mM MgCl2、50 mM NaCl、1 mM 二硫苏糖醇(缓冲液NEB2)存在下,将寡核苷酸 EarV2-Random和EarV2-Lower加热至90℃并在5分钟内冷却至10℃。在加入所有4种DNA核苷酸和E. coli的DNA聚合酶的Klenow片段后,通过在室温下培养15分钟合成作为结果形成的部分双链DNA碎片以完成双链。然后,使用寡核苷酸 EarV2-UpAmp和EarV2-LoAmp将反应混合物引入PCR反应中,以便扩增获得的双链。由于在每种情况下扩增的寡核苷酸 EarV2-UpAmp和EarV2-LoAmp与新形成的双链的重叠仅为12bp,未使用PRC试剂盒的热稳定聚合酶而是使用E. coli的DNA 聚合酶的Klenow片段进行最初的两个扩增循环,以便实现用作引物的寡核苷酸 EarV2-UpAmp和EarV2-LoAmp的结合。为此,将起始反应升至94℃保持1分钟,随后置于冰上。然后,加入1 µL的DNA 聚合酶 (Klenow 片段) ,在18℃下培养3分钟,在25℃下培养1分钟。然后,将起始反应再次升至94℃保持1分钟,随后置于冰上。加入1 µL的DNA 聚合酶 (Klenow 片段) ,在18℃下培养3分钟,在25℃下培养1分钟。仅在此时在PCR机器中进行下列扩增程序(20 循环):在94℃下1分钟,在52℃下1分钟,在72℃下1分钟。
在PCR扩增后,通过柱色谱(PCR Purification Kit,Qiagen)提纯反应并使用EarI剪切。随后在琼脂糖凝胶 (4% 琼脂糖) 上分离反应混合物,从凝胶上分离所需的64 bp 片段(SEQ ID 33)并提纯。
使用EcoO109I剪切实施例1所述的载体 pAI3B-KOZ65-U,随后根据生产商的说明 (New England Biolabs) 使用碱性磷酸酶脱磷酸以防止载体的再环化(Rezirkularisierung)。随后在16℃下以5:1的比例将64 bp 片段和使用EcoO109I线性化的脱磷酸载体连接过夜。64 bp 片段可以以相对于载体的所需方向而非其他方向连接于载体,从这个意义上来说,EcoO109I和EarI形成的5’ 突出端是适合的。因此排除具有错误方向的插入的连接产物,而非在每种情况下方向正确的多聚体的形成。然而,这种基因不会导致功能性sPQQGDH蛋白的表达,因为阅读框中发生了移位。将得到的连接产物转化到E. coli-DH5α中。由最初的转化混合物,将1/25、4/25和20/25各自涂布到20 cm x 20 cm大小的琼脂板上,以便通过至少一次稀释,获得每板约1000-5000个菌落的菌落密度。
在该实施例中概述了在每种情况下克隆表达具有4种其他氨基酸的sPQQGDH 变体的库的生产。以相同的方式实施具有其他长度的插入的库的生产。
实施例3 – 筛选变体-库
从根据实施例2所述的方法生产的库中选取合适的变体,借助自动菌落采集器QPix (Fa. Genetix) 从具有总共约4000个菌落的两个琼脂板中采集2200个菌落,转移至微量滴定板 (MTPs)中,在每种情况下,所述微量滴定板每孔(Well)具有含有100 µg/mL氨苄西林的200 µL的LB 培养基。使用原始菌株E. coli-DH5α::pAI3B-KOZ65的培养物另外接种4个MTP上的总共8个孔。使用透气薄膜(Airpore Sheets,Qiagen)密封MTP,在37℃下振摇5小时。随后,将50 µL各个单独培养物转移到同样在MTP中的新的培养容器中,在新的培养容器中存在含有氨苄西林的150 µL LB 培养基。为此,使用平行执行96次移液操作的移液机器人。该培养基另外接受200 ng/mL无水四环素,以便诱导sPQQGDH基因的表达。这些MTP在28℃下振摇过夜。
为了能够评价大量菌落的酶活性,使用不需要破裂细胞的试验:在通过加入PQQ激活酶之后,由于在二氯酚靛酚 (DCPIP)还原后的脱色作用,可以直接追踪糖的氧化。为了进行筛选,从所有孔中,将每个孔的40 µL诱导培养物与60 µL 葡萄糖测试溶液和另外的40 µL与60 µL 麦芽糖测试溶液混合,其中一个MTP仅含有葡萄糖测试溶液或仅含有麦芽糖测试溶液。同样使用移液机器人进行该操作。以这种方式,能够将各个单独的培养物分配到所有培养板和测量板上连续不变的位置。葡萄糖测试溶液含有下列组分(括号内:起始测量溶液中的最终浓度):50 mM 葡萄糖(30 mM),750 µM DCPIP (450 µM),1.5 µM PQQ (0.9 µM),1 mM CaCl2 (0.6
mM),0.1% NaN3
(0.06%),62.5 ppm (37.5 ppm) 硅酮消泡剂 (Fluka # 85390),50 mM 三(羟甲基)氨基甲烷-盐酸盐(TRIS-HCl),pH 7.6 (30 mM)。在麦芽糖测试溶液的情况中,不存在葡萄糖,但存在相同浓度的麦芽糖。
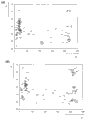
对于每个具有培养物的MTP,以这种方式生成两块测量板,一块用于葡萄糖,另一块用于麦芽糖。对于每块测量板,在培养物和测试溶液混合后测定 (Eppendorf-Biophotometer)的不同的时间点在605nm的波长下的光密度:1分钟后,7分钟后,12分钟后和18分钟后。以这种方式,对于每个最初分离的菌落,能够记录葡萄糖作为底物的酶动力学和麦芽糖作为底物的酶动力学。由此,能够确定克隆与作为底物的葡萄糖彼此间的相对活性以及每种单独的克隆对葡萄糖和麦芽糖的活性比。图3显示对于由筛选在位置Q168和L169之间具有5个其他氨基酸的库得到的MTP 13和22的这种测量的结果。很明显,约2/3的测试菌落具有小于50个相对单位的葡萄糖转化率,低于测试的读出准确度。在这些菌落中,没有检测到sPQQGDH的显著表达,其导致非常低的培养物生长、差的sPQQGDH基因诱导,不生产的双插入或氨基酸组合的插入,其不再使形成的蛋白具有酶活性。与葡萄糖相比,绝大部分数量的剩余菌落显示对麦芽糖的更低活性。如图3所示,与原始克隆 E. coli-DH5α::pAI3B-KOZ65相比,初步筛选的结果显示相似的许多变体的高转化率。然而,应当始终检查相对活性,因为在大于某一转化率时,设计用于非常多的单个测定的这个实验不能进行区分。与KOZ65的sPQQGDH相比,变体的sPQQGDH通常具有比由所示筛选所预期的更低的转化率。
实施例4 – 单个菌落的精细筛选
实施例3中所述的筛选用于选择188个菌落,其中对葡萄糖的活性和对葡萄糖或麦芽糖的活性的比例都显示特别有前景,如实施例3所述,将这些菌落与原始菌株E. coli-DH5α::pAI3B-KOZ65的培养物一起安排在两个新的MTP上,开始在37℃下生长,然后在28℃下诱导过夜。为了进行诱导,每个MTP设置两块板,以便得到足够的进行筛选实验的培养物体积。在诱导后,将各自约为200 µL的平行生长的培养物合并并混和。在每种情况下,由每个这些诱导的培养物中,在各种情况下转移40 µL到测量板中,其中在每种情况下存在60 µL实施例3中所述的测试溶液,但是具有下列糖:G – 50 mM 葡萄糖,M – 50 mM 麦芽糖,GM – 50 mM 葡萄糖和50 mM 麦芽糖,GGal – 50 mM 葡萄糖和50 mM 半乳糖,或GXyl – 50 mM 葡萄糖和50 mM 木糖。在培养物和测试溶液混合30分钟后以30秒的间隔迅速测量这些板,以便获得各种单个培养物的动力学。由这样获得的数据,可以估计每个培养物的葡萄糖和麦芽糖单独的转化率、对葡萄糖和麦芽糖活性的比例,以及特别地,麦芽糖、半乳糖和木糖对葡萄糖转化的影响(干扰)。在图4中,显示了选择用于实施例3中所述筛选的菌落(第2次筛选)的这种随访研究的结果。
第一次筛选的MTP 13的两个菌落(13-G3和13-B2;在图3和4中各自使用矩形标记)通过实施例表明,具有有前景的麦芽糖活性对葡萄糖活性的比例的菌落(图3)能够显示强烈地偏离干扰行为:13-G3经历麦芽糖的负干扰;13-B2经历正干扰。
根据两次所述的筛选,选择总共28个菌落用于进一步的研究。
实施例5 –表达变体形式的sPQQGDH的菌株的生长
首先,由四种所选的菌落的每种如下生产预培养物,所述菌落来自在每种情况下在位置Q168和L169之间具有3个其他氨基酸的库:使用所检查的菌落接种2 mL TB 培养基(含有100 μg/mL 氨苄西林),在37℃和225rpm下振摇过夜。对于主要培养物,使用完全生长的预培养物接种50 mL TB 培养基(100 μg/mL 氨苄西林)。在37℃和225rpm下振摇培养物,直至达到在605 nm (OD605)下的光密度约为1。然后,通过加入无水四环素 (AHT) 菌株溶液诱导酶的表达。为此,将诱导物溶解于DMF中。AHT在培养物中的最终浓度为0.2 μg/mL;诱导在27℃和225rpm下进行24小时。
在4℃下通过在3220 x g下将主要培养物离心15分钟收集细胞。根据量,将片状沉淀细胞每次再混悬于用量为10–20 mL的pH 7.6的50 mM 3-吗啉代丙烷磺酸缓冲液(MOPS)中和2.5 mM CaCl2中,通过超声溶解处理,直至观察到显著澄清的细胞混悬液。在4℃下将细胞碎片和任何存在的包涵体在48,745 x g下离心10分钟。
实施例6 – 变体sPQQGDH 的提纯
使用柱缓冲液(10 mM MOPS,pH 7.6 + 2.5 mM CaCl2)以1:5将实施例5的上清液稀释至10或50 mL,通过阳离子交换剂(Toyopearl CM-650M,Fa. TOSOH BIOSEP GmbH)进行色谱拆分。为此,使用具有约16 mL柱床的10 x 1.42 cm柱;流速为4 mL/分。使用10倍柱体积的缓冲液平衡柱;然后,应用样品。使用4 倍柱体积的缓冲液洗涤柱;随后,通过线性盐梯度从0-0.4 M 在柱缓冲液中的NaCl洗脱sPQQGDH,以1或3 mL的级分收集洗脱物。在这种条件下洗脱的sPQQGDH事实上为同质蛋白质,所以不需要进一步地提纯。为了测定蛋白质,测定溶液的OD280。使用纯净的KOZ65的sPQQGDH蛋白质,先前通过重量分析已经确定,1 mg/mL sPQQGDH溶液(PQQ-游离的酶蛋白)的OD280为1.27。
实施例7 – 确定sPQQGDH 变体的酶活性
为了精确地测量酶活性,使用下列试验。在每种情况下制备所研究的酶溶液在下列缓冲液中的稀释系列:50 mM 1,4-哌嗪-二-(2-乙磺酸) (PIPES),pH 6.5,0.1% Triton X-100,1 mM CaCl2,0.1% 牛血清白蛋白 (BSA)和3 µM PQQ。糖用作底物,分别制备电子传递体N-甲基phenazoniummethasulfat
(PMS) 和检测试剂氯化硝基四氮唑蓝 (NBT):将11.7 mL的pH 6.5的50 mM PIPES和2% Triton X-100在重蒸馏的(bidest.)水中的溶液与450 µL 0.9 M D-葡萄糖、450 µL 6 mM PMS和450 µL 6.6 mM NBT(所有物质也溶解于重蒸馏的水中)混合,在25℃下保持在暗处,在1小时内用完。当测量对除葡萄糖外的糖的酶活性时,这些糖,而不是葡萄糖,以0.9 M的浓度使用。单个反应物在测试溶液中的最终浓度为:30 mM 糖底物,200 µM PMS,和220 µM NBT。将(725 µL)预热的测试溶液与25 µL稀释的酶溶液在试管中混合,在570nm和25℃下在3分钟内追踪甲簪的发展。1 µmol葡萄糖转化得到0.5 µmol甲簪,其摩尔消光系数为ε = 40,200 M-1cm-1。因此,样品中的酶浓度可以通过下列关系确定:1 U/mL sPQQGDH对应1.493 min-1的OD570中的改变。单个样品的稀释系列是必需的,因为其中发现值低于0.05 U/mL或大于0.7 U/mL的测量必须抛弃,因为不能保证在这个范围外的足够测量线性。然后计算原始样品的浓度,考虑所用的稀释水平。结果显示于下列表2中。
表 2:
实施例8 – 测定插入序列
为了确定在感兴趣的变体中在位置Q168和L169之间插入的实际氨基酸,根据生产商的说明,借助QiaPrep 质粒分离试剂盒(Qiagen,Hilden) 由所关注的菌株制备质粒DNA,使用用作引物的寡核苷酸 4472-US1 (5’-CACCGTTAAAGCTTGGATTATC-3’;SEQ ID 34) 和4831-DS1 (5’-CCTTCATCGAAAGACCATCAG-3’;SEQ ID 35)由两个方向确定插入位点的DNA 序列。4472-US1序列的3’端位于插入位点的58 bp上游;4831-DS1序列的3’端位于插入位点的82 bp下游。序列分析的结果列于下列表3中。
表 3:
为了进行定向,还显示了侧翼氨基酸168Q和172L (先前为169L)以及它们的密码子。
实施例9 – 确定除葡萄糖外的糖的干扰
虽然在起始测试溶液中30 mM的底物浓度通常用于测定sPQQGDH的比活性,但更低浓度的所讨论的糖对于确定干扰是能察觉的;在可能的后续应用中大约以数量级出现。葡萄糖在血液中的生理浓度为约100 mg/dL,相当于约5.6 mM。由此原因,使用100 mg/dL的葡萄糖浓度和50-250 mg/dL的干扰糖进行下文描述的实验。然而在某些情况中, 还研究了其他浓度和两种糖相互之间的比例。
在图5中,显示了5种不同变体的干扰测定的结果。5381-06-C12、5381-05-C4和5381-07-F10来源于在位置Q168和L169之间具有4个其他氨基酸的库;5152-20-A7和5386-13-F10来源于在位置Q168和L169之间具有5个其他氨基酸的库。在所有情况中,仍然能检测到所测试糖仅有的较小影响:在麦芽糖的情况中,使用所有克隆的测量受影响不超过8%,即使是在250mg/dL 麦芽糖 (与葡萄糖相比,1.3-倍摩尔量)下。在半乳糖的情况下,干扰最大为16%,而在木糖的情况下,干扰最大为14%。
实施例10 – 使用KOZ65和克隆 5152-20-A7进行的不同糖的干扰的比较
为了对没有插入突变的来自KOZ65的sPQQGDH之间进行直接的比较,在葡萄糖和糖麦芽糖、半乳糖、木糖、乳糖、纤维二糖或甘露糖的混合物中研究纯净的来自克隆 5152-20-A7的变体sPQQGDH和纯净的来自KOZ65的酶。对于每个测量点,一式四份进行记录。图6显示麦芽糖、纤维二糖和乳糖对KOZ65的原始酶的强干扰在通过变体5152-20-A7表达的酶中被抑制。更特别地,在约1等摩尔量下麦芽糖的干扰(100 mg/dL 葡萄糖,含有200 mg/dL 麦芽糖)从KOZ65的63%下降至5152-20-A7的6%;使用乳糖,干扰不再是显著的。
实施例11 – 不同库的变体的比较
为了显示用于生产具有改善的底物特异性的sPQQGDH 变体的根据本发明的方法的一般有效性,建立并研究在KOZ65的野生型sPQQGDH的位置Q168和L169之间具有3个、4个和5个其他氨基酸的库,如先前实施例中所述的那样。为了进行比较,还建立并研究在位置Q168和L169之间仅具有2个其他氨基酸的库。然而,发现仅有10个底物特异性具有轻微改进的克隆(对麦芽糖和葡萄糖的活性比在25-75%之间)。仅有1个克隆(5156-13-G12;参见表4)具有增强的底物特异性,但具有差的对葡萄糖的比活性。然而正如由下表4明显看出,所有具有3-5个其他氨基酸的库含有,显示比野生型明显更高的底物特异性的克隆。
表4:
(n.d.: 未测定)
图1 显示用于合成的DNA片段的插入的载体的生产。显示了用于生产来自KOZ65的sPQQGDH基因 (1a)的两个片段(PCR 1,PCR 2)的引物的位置和方向 (1b中粗的,黑色箭头)以及其他氨基酸随后插入的位置(1a和1b中的"IP")。图1c图解式地显示了编码序列(其已经使用XbaI和EcoO109I进行剪切并提纯)的上部的扩增子、编码序列(其已经使用EcoO109I和BfuAI进行剪切并提纯)中部的扩增子的载体 pAI3B-KOZ65-U (1-2)、3567 bp长的载体片断(其已经由原始载体pAI3B-KOZ65 (1-1) 使用XbaI和BfuAI进行剪切并提纯)的装配。完成的载体 pAI3B-KOZ65-U (1-2)显示于图1e中。由图1d可以看出,当没有发生插入时,pAI3B-KOZ65-U中的sPQQGDH翻译终止在EcoO109I 限制位点之后4个氨基酸残基。
图2显示由文中所述的寡核苷酸进行合成的、双链DNA片段的制备(在图中显示为箭头,箭头方向在每种情况下表示序列的3’端)。在下部,显示了寡核苷酸 EarV2-Random和EarV2-Lower。在第一步中,由这些重叠单链制造62 bp长的初级产物。借助寡核苷酸 EarV2-UpAmp和EarV2-LoAmp,在第二步中由其生产的是97 bp长的PCR产物,其DNA 序列显示于上部。其编码的蛋白质序列见于图2的下缘。在DNA序列中,显示了两个EarI 限制位点的位置。将使用EarI 由PCR产物释放的64 bp长的片段连接于使用EcoO109I开放的载体。
图3 显示使用两个微量滴定板 (a和b)的样本(Beispiel)筛选变体sPQQGDH 克隆的库的数据概要。横座标(X-轴)给出了MTP的每个菌落以任意相对单位计的被该菌落转化的葡萄糖的近似率。在纵座标(Y-轴)上,绘制麦芽糖转化和葡萄糖转化的商(Quotient)。对于每个菌落,这能够评价菌落是否能够转化葡萄糖,并且如果是这样,菌落是否可以比野生型KOZ65更好地区分葡萄糖和麦芽糖。作为每个菌落的象征,使用其在各个微量滴定板上的位置,A1–H12。阳性对照KOZ65用箭头表示。
图4显示由实施例3中所述的筛选选择的菌落的结果。考虑三种参数:对葡萄糖的转化率(未显示在该图中)应当尽可能的高;横座标(X-轴)上绘制的干扰(这里,麦芽糖是干扰糖),以及纵座标(Y-轴) 上绘制的麦芽糖转化与葡萄糖转化的比例应当尽可能地接近0。为了进行比较,以相同的方式标记图3和4中的一些克隆。
图5显示不同克隆与麦芽糖 (a) 、半乳糖 (b)、和木糖 (c)的干扰研究。显示了5种纯净的变体sPQQGDH制品对葡萄糖和三种其他糖的混合物的行为。纵座标(Y-轴)绘制了确定的起始测试溶液中100 mg/dL 葡萄糖不与其它糖的混合物的干扰,以及与50、100、150、200或250 mg/dL (X-轴)的各种干扰糖的混合物的干扰。每个测试点一式四份进行测定。
图6 显示KOZ65 (左)和克隆 5152-20-A7 (右)对各种糖的干扰研究。对于纤维二糖(a)、半乳糖 (b)、乳糖(c)、麦芽糖 (d)、甘露糖(e)和木糖 (f),在纵座标(Y-轴)上绘制在100 mg/dL 葡萄糖存在下(在X-轴上的抑制剂浓度[mg/dL])这些糖在各种浓度下如何强烈地影响KOZ65的sPQQGDH或5152-20-A7的活性。对于干扰糖,每个测量系列还包括不含这种糖的测量,并且这种测量,像该系列的所有其他测量一样,是基于由所有不含干扰糖的测量平均得到的葡萄糖值。由于酶试验的测量不准确性,单个图形相应地不一定从零起始,虽然理论上是这样。
序列表
<110> 拜耳技术服务 GmbH
<120> 具有改善的底物特异性的PQQ-依赖性葡萄糖脱氢酶的新变体
<130> AFR
<160>
35
<170>
PatentIn version 3.3
<210> 1
<211> 3
<212> PRT
<213> 人工的
<220>
<223> 插入
<400> 1
Ala Tyr Gln
1
<210> 2
<211> 3
<212> PRT
<213> 人工的
<220>
<223> 插入
<400> 2
Ala Trp Leu
1
<210> 3
<211> 3
<212> PRT
<213> 人工的
<220>
<223> 插入
<400> 3
Ala Phe Val
1
<210> 4
<211> 3
<212> PRT
<213> 人工的
<220>
<223> 插入
<400> 4
Gly Tyr Ile
1
<210> 5
<211> 3
<212> PRT
<213> 人工的
<220>
<223> 插入
<400> 5
Ala Tyr Val
1
<210> 6
<211> 3
<212> PRT
<213> 人工的
<220>
<223> 插入
<400> 6
Ala Phe Gln
1
<210> 7
<211> 4
<212> PRT
<213> 人工的
<220>
<223> 插入
<400> 7
Ala Gly Arg Met
1
<210> 8
<211> 4
<212> PRT
<213> 人工的
<220>
<223> 插入
<400> 8
Gly Leu Ala Val
1
<210> 9
<211> 5
<212> PRT
<213> 人工的
<220>
<223> 插入
<400> 9
Met Gly Arg Phe Leu
1 5
<210> 10
<211> 5
<212> PRT
<213> 人工的
<220>
<223> 插入
<400> 10
Val Ser Thr Phe Phe
1
5
<210> 11
<211> 5
<212> PRT
<213> 人工的
<220>
<223> 插入
<400> 11
Val Ser Lys Asn His
1
5
<210> 12
<211> 5
<212> PRT
<213> 人工的
<220>
<223> 插入
<400> 12
Ser Ser Arg Asn His
1
5
<210> 13
<211> 5
<212> PRT
<213> 人工的
<220>
<223> 插入
<400> 13
Ser Gly Arg Ile Leu
1
5
<210> 14
<211> 5
<212> PRT
<213> 人工的
<220>
<223> 插入
<400> 14
Val Gly Arg Leu Thr
1
5
<210> 15
<211> 5
<212> PRT
<213> 人工的
<220>
<223> 插入
<400> 15
Ala Glu Arg Asn Tyr
1
5
<210> 16
<211> 5
<212> PRT
<213> 人工的
<220>
<223> 插入
<400> 16
Met Glu Ser His Asn
1
5
<210> 17
<211> 5
<212> PRT
<213> 人工的
<220>
<223> 插入
<400> 17
Val Gly His Val Thr
1
5
<210> 18
<211> 5
<212> PRT
<213> 人工的
<220>
<223> 插入
<400> 18
Val Gly Arg Tyr Gln
1 5
<210> 19
<211> 42
<212> DNA
<213> 人工的
<220>
<223> 合成的寡核苷酸
<400> 19
tggtaggtct caaatgaata aacatttatt ggctaaaatt ac 42
<210> 20
<211> 32
<212> DNA
<213> 人工的
<220>
<223> 合成的寡核苷酸
<400> 20
ttcagctctg agctttatat gtaaatctaa tc 32
<210> 21
<211>
4560
<212> DNA
<213> 人工的
<220>
<223> 表达质粒
<220>
<221> CDS
<222>
(139)..(1581)
<220>
<221>
sig_肽
<222>
(139)..(213)
<220>
<221>
mat_肽
<222>
(214)..(1581)
<400> 21
ccatcgaatg gccagatgat taattcctaa tttttgttga cactctatca
ttgatagagt 60
tattttacca ctccctatca gtgatagaga aaagtgaaat gaatagttcg
acaaaaatct 120
agataacgag ggcaaaaa atg aat aaa cat tta ttg gct aaa
att act tta 171
Met Asn Lys His Leu Leu Ala Lys Ile Thr Leu
-25 -20 -15
tta ggt gct gct cag cta ctt acg ctc aat tca gca ttt
gct gat gtc 219
Leu Gly Ala Ala Gln Leu Leu Thr Leu Asn Ser Ala Phe
Ala Asp Val
-10 -5 -1 1
cct ctt aca cca tct caa ttt gct aaa gcg aaa aca gaa
agc ttt gat 267
Pro Leu Thr Pro Ser Gln Phe Ala Lys Ala Lys Thr Glu
Ser Phe Asp
5 10 15
aag aaa gtt ctt cta tct aat tta aat aag cca cat gct
ttg ttg tgg 315
Lys Lys Val Leu Leu Ser Asn Leu Asn Lys Pro His Ala
Leu Leu Trp
20 25 30
gga ccg gat aat caa att tgg tta acg gag cgg gca aca
ggt aag att 363
Gly Pro Asp Asn Gln Ile Trp Leu Thr Glu Arg Ala Thr
Gly Lys Ile
35
40 45 50
cta aga gtt aat cca gag tcg ggc agt gta aaa aca gtt
ttt cag gtt 411
Leu Arg Val Asn Pro Glu Ser Gly Ser Val Lys Thr Val
Phe Gln Val
55 60 65
cct gag att gta aat gat gct gat gga caa aac ggt tta
ttg ggt ttt 459
Pro Glu Ile Val Asn Asp Ala Asp Gly Gln Asn Gly Leu
Leu Gly Phe
70 75 80
gcc ttt cat cct gac ttt aaa aat aat cct tat atc tat
gtt tca ggt 507
Ala Phe His Pro Asp Phe Lys Asn Asn Pro Tyr Ile Tyr
Val Ser Gly
85 90 95
aca ttt aaa aat ccg aaa tct aca gat aaa gaa tta ccg
aat caa act 555
Thr Phe Lys Asn Pro Lys Ser Thr Asp Lys Glu Leu Pro
Asn Gln Thr
100 105 110
att atc cgt cga tat acc tat aac aag gca aca gat acc
ctt gag aaa 603
Ile Ile Arg Arg Tyr Thr Tyr Asn Lys Ala Thr Asp Thr
Leu Glu Lys
115
120 125 130
cca gta gat tta ttg gca gga tta cct tca tcg aaa gac
cat cag tcg 651
Pro Val Asp Leu Leu Ala Gly Leu Pro Ser Ser Lys Asp
His Gln Ser
135 140 145
ggt cgt ctt gtg att ggt cca gac caa aag att tac tat
acg att ggt 699
Gly Arg Leu Val Ile Gly Pro Asp Gln Lys Ile Tyr Tyr
Thr Ile Gly
150 155 160
gat cag gga cgt aac cag ctg gct tat tta ttc tta cca
aat caa gca 747
Asp Gln Gly Arg Asn Gln Leu Ala Tyr Leu Phe Leu Pro
Asn Gln Ala
165 170 175
cag cat acg ccg act caa cag gaa ctg agc ggc aaa gac
tat cat acc 795
Gln His Thr Pro Thr Gln Gln Glu Leu Ser Gly Lys Asp
Tyr His Thr
180 185 190
tat atg ggt aaa gta ttg cgc tta aat ctg gat gga agt
att cca aaa 843
Tyr Met Gly Lys Val Leu Arg Leu Asn Leu Asp Gly Ser
Ile Pro Lys
195
200 205 210
gat aat cca agc ttt aac ggt gta att agc cat att tat
acg ctc ggt 891
Asp Asn Pro Ser Phe Asn Gly Val Ile Ser His Ile Tyr
Thr Leu Gly
215 220 225
cat cgt aac cca cag ggc ttg gca ttt act cca aat ggt
aaa ctg ttg 939
His Arg Asn Pro Gln Gly Leu Ala Phe Thr Pro Asn Gly
Lys Leu Leu
230 235 240
caa tct gaa cag ggt cca aac tct gat gat gaa att aac
ctc att gtc 987
Gln Ser Glu Gln Gly Pro Asn Ser Asp Asp Glu Ile Asn
Leu Ile Val
245 250 255
aaa ggt ggt aac tat ggc tgg cca aat gta gcg ggt tat
aaa gat gat 1035
Lys Gly Gly Asn Tyr Gly Trp Pro Asn Val Ala Gly Tyr
Lys Asp Asp
260 265 270
agt ggt tat gcc tat gca aat tat tcg gca gca agc aat
aaa gca caa 1083
Ser Gly Tyr Ala Tyr Ala Asn Tyr Ser Ala Ala Ser Asn
Lys Ala Gln
275
280 285 290
att aaa gat tta gga caa aat ggt tta aaa gtg gcg gca
ggt gta cct 1131
Ile Lys Asp Leu Gly Gln Asn Gly Leu Lys Val Ala Ala
Gly Val Pro
295 300 305
gtg atg aaa gag tct gaa tgg act ggt aaa aac ttt gta
ccg ccg tta 1179
Val Met Lys Glu Ser Glu Trp Thr Gly Lys Asn Phe Val
Pro Pro Leu
310 315 320
aaa act tta tat acc gtc caa gat acc tat aac tat aat
gac cca act 1227
Lys Thr Leu Tyr Thr Val Gln Asp Thr Tyr Asn Tyr Asn
Asp Pro Thr
325 330 335
tgt ggg gat atg acc tac att tgc tgg cca acg gtt gcg
ccg tca tct 1275
Cys Gly Asp Met Thr Tyr Ile Cys Trp Pro Thr Val Ala
Pro Ser Ser
340 345 350
gct tat gtc tat aag gga ggc aaa aaa gca att tct ggt
tgg gaa aat 1323
Ala Tyr Val Tyr Lys Gly Gly Lys Lys Ala Ile Ser Gly
Trp Glu Asn
355
360 365 370
aca tta ttg gtt cca tct tta aag cgc ggt gtt att ttc
cgt att aag 1371
Thr Leu Leu Val Pro Ser Leu Lys Arg Gly Val Ile Phe
Arg Ile Lys
375 380 385
cta gat cca act tac agt act act tat gat gat gct gtg
ccg atg ttt 1419
Leu Asp Pro Thr Tyr Ser Thr Thr Tyr Asp Asp Ala Val
Pro Met Phe
390 395 400
aag agc aac aat cgt tat cgt gac gtg att gca agt cca
gat ggg aat 1467
Lys Ser Asn Asn Arg Tyr Arg Asp Val Ile Ala Ser Pro
Asp Gly Asn
405 410 415
gtt tta tat gta ttg act gat act tcc gga aat gtc caa
aaa gat gat 1515
Val Leu Tyr Val Leu Thr Asp Thr Ser Gly Asn Val Gln
Lys Asp Asp
420 425 430
ggt tct gta acg aat aca tta gaa aac cca gga tct ctg
att aga ttt 1563
Gly Ser Val Thr Asn Thr Leu Glu Asn Pro Gly Ser Leu Ile
Arg Phe
435
440 445 450
aca tat aaa gct cag agc tgaaagcttg acctgtgaag
tgaaaaatgg 1611
Thr Tyr Lys Ala Gln Ser
455
cgcacattgt gcgacatttt ttttgtctgc cgtttaccgc tactgcgtca
cggatctcca 1671
cgcgccctgt agcggcgcat taagcgcggc gggtgtggtg gttacgcgca
gcgtgaccgc 1731
tacacttgcc agcgccctag cgcccgctcc tttcgctttc ttcccttcct
ttctcgccac 1791
gttcgccggc tttccccgtc aagctctaaa tcgggggctc cctttagggt
tccgatttag 1851
tgctttacgg cacctcgacc ccaaaaaact tgattagggt gatggttcac
gtagtgggcc 1911
atcgccctga tagacggttt ttcgcccttt gacgttggag tccacgttct
ttaatagtgg 1971
actcttgttc caaactggaa caacactcaa ccctatctcg gtctattctt
ttgatttata 2031
agggattttg ccgatttcgg cctattggtt aaaaaatgag ctgatttaac
aaaaatttaa 2091
cgcgaatttt aacaaaatat taacgcttac aatttcaggt ggcacttttc
ggggaaatgt 2151
gcgcggaacc cctatttgtt tatttttcta aatacattca aatatgtatc
cgctcatgag 2211
acaataaccc tgataaatgc ttcaataata ttgaaaaagg aagagtatga
gtattcaaca 2271
tttccgtgtc gcccttattc ccttttttgc ggcattttgc cttcctgttt
ttgctcaccc 2331
agaaacgctg gtgaaagtaa aagatgctga agatcagttg ggtgcacgag
tgggttacat 2391
cgaactggat ctcaacagcg gtaagatcct tgagagtttt cgccccgaag
aacgttttcc 2451
aatgatgagc acttttaaag ttctgctatg tggcgcggta ttatcccgta
ttgacgccgg 2511
gcaagagcaa ctcggtcgcc gcatacacta ttctcagaat gacttggttg
agtactcacc 2571
agtcacagaa aagcatctta cggatggcat gacagtaaga gaattatgca
gtgctgccat 2631
aaccatgagt gataacactg cggccaactt acttctgaca acgatcggag
gaccgaagga 2691
gctaaccgct tttttgcaca acatggggga tcatgtaact cgccttgatc
gttgggaacc 2751
ggagctgaat gaagccatac caaacgacga gcgtgacacc acgatgcctg
tagcaatggc 2811
aacaacgttg cgcaaactat taactggcga actacttact ctagcttccc
ggcaacaatt 2871
gatagactgg atggaggcgg ataaagttgc aggaccactt ctgcgctcgg
cccttccggc 2931
tggctggttt attgctgata aatctggagc cggtgagcgt ggctctcgcg
gtatcattgc 2991
agcactgggg ccagatggta agccctcccg tatcgtagtt atctacacga
cggggagtca 3051
ggcaactatg gatgaacgaa atagacagat cgctgagata ggtgcctcac
tgattaagca 3111
ttggtaggaa ttaatgatgt ctcgtttaga taaaagtaaa gtgattaaca
gcgcattaga 3171
gctgcttaat gaggtcggaa tcgaaggttt aacaacccgt aaactcgccc
agaagctagg 3231
tgtagagcag cctacattgt attggcatgt aaaaaataag cgggctttgc
tcgacgcctt 3291
agccattgag atgttagata ggcaccatac tcacttttgc cctttagaag
gggaaagctg 3351
gcaagatttt ttacgtaata acgctaaaag ttttagatgt gctttactaa
gtcatcgcga 3411
tggagcaaaa gtacatttag gtacacggcc tacagaaaaa cagtatgaaa
ctctcgaaaa 3471
tcaattagcc tttttatgcc aacaaggttt ttcactagag aatgcattat
atgcactcag 3531
cgcagtgggg cattttactt taggttgcgt attggaagat caagagcatc
aagtcgctaa 3591
agaagaaagg gaaacaccta ctactgatag tatgccgcca ttattacgac
aagctatcga 3651
attatttgat caccaaggtg cagagccagc cttcttattc ggccttgaat
tgatcatatg 3711
cggattagaa aaacaactta aatgtgaaag tgggtcttaa aagcagcata
acctttttcc 3771
gtgatggtaa cttcactagt ttaaaaggat ctaggtgaag atcctttttg
ataatctcat 3831
gaccaaaatc ccttaacgtg agttttcgtt ccactgagcg tcagaccccg
tagaaaagat 3891
caaaggatct tcttgagatc ctttttttct gcgcgtaatc tgctgcttgc
aaacaaaaaa 3951
accaccgcta ccagcggtgg tttgtttgcc ggatcaagag ctaccaactc
tttttccgaa 4011
ggtaactggc ttcagcagag cgcagatacc aaatactgtc cttctagtgt
agccgtagtt 4071
aggccaccac ttcaagaact ctgtagcacc gcctacatac ctcgctctgc
taatcctgtt 4131
accagtggct gctgccagtg gcgataagtc gtgtcttacc gggttggact
caagacgata 4191
gttaccggat aaggcgcagc ggtcgggctg aacggggggt tcgtgcacac
agcccagctt 4251
ggagcgaacg acctacaccg aactgagata cctacagcgt gagctatgag
aaagcgccac 4311
gcttcccgaa gggagaaagg cggacaggta tccggtaagc ggcagggtcg
gaacaggaga 4371
gcgcacgagg gagcttccag ggggaaacgc ctggtatctt tatagtcctg
tcgggtttcg 4431
ccacctctga cttgagcgtc gatttttgtg atgctcgtca ggggggcgga
gcctatggaa 4491
aaacgccagc aacgcggcct ttttacggtt cctggccttt tgctggcctt
ttgctcacat 4551
gacccgaca
4560
<210> 22
<211> 481
<212> PRT
<213> 人工的
<220>
<223> 合成构建体
<400> 22
Met Asn Lys His Leu Leu Ala Lys Ile Thr Leu Leu Gly
Ala Ala Gln
-25
-20 -15 -10
Leu Leu Thr Leu Asn Ser Ala Phe Ala Asp Val Pro Leu
Thr Pro Ser
-5 -1
1 5
Gln Phe Ala Lys Ala Lys Thr Glu Ser Phe Asp Lys Lys
Val Leu Leu
10 15 20
Ser Asn Leu Asn Lys Pro His Ala Leu Leu Trp Gly Pro
Asp Asn Gln
25 30 35
Ile Trp Leu Thr Glu Arg Ala Thr Gly Lys Ile Leu Arg
Val Asn Pro
40
45 50 55
Glu Ser Gly Ser Val Lys Thr Val Phe Gln Val Pro Glu
Ile Val Asn
60 65 70
Asp Ala Asp Gly Gln Asn Gly Leu Leu Gly Phe Ala Phe
His Pro Asp
75 80 85
Phe Lys Asn Asn Pro Tyr Ile Tyr Val Ser Gly Thr Phe
Lys Asn Pro
90 95 100
Lys Ser Thr Asp Lys Glu Leu Pro Asn Gln Thr Ile Ile
Arg Arg Tyr
105 110 115
Thr Tyr Asn Lys Ala Thr Asp Thr Leu Glu Lys Pro Val
Asp Leu Leu
120
125 130 135
Ala Gly Leu Pro Ser Ser Lys Asp His Gln Ser Gly Arg
Leu Val Ile
140 145 150
Gly Pro Asp Gln Lys Ile Tyr Tyr Thr Ile Gly Asp Gln
Gly Arg Asn
155 160 165
Gln Leu Ala Tyr Leu Phe Leu Pro Asn Gln Ala Gln His
Thr Pro Thr
170 175 180
Gln Gln Glu Leu Ser Gly Lys Asp Tyr His Thr Tyr Met
Gly Lys Val
185 190 195
Leu Arg Leu Asn Leu Asp Gly Ser Ile Pro Lys Asp Asn
Pro Ser Phe
200
205 210 215
Asn Gly Val Ile Ser His Ile Tyr Thr Leu Gly His Arg
Asn Pro Gln
220 225 230
Gly Leu Ala Phe Thr Pro Asn Gly Lys Leu Leu Gln Ser
Glu Gln Gly
235 240 245
Pro Asn Ser Asp Asp Glu Ile Asn Leu Ile Val Lys Gly
Gly Asn Tyr
250 255 260
Gly Trp Pro Asn Val Ala Gly Tyr Lys Asp Asp Ser Gly
Tyr Ala Tyr
265 270 275
Ala Asn Tyr Ser Ala Ala Ser Asn Lys Ala Gln Ile Lys
Asp Leu Gly
280
285 290 295
Gln Asn Gly Leu Lys Val Ala Ala Gly Val Pro Val Met
Lys Glu Ser
300 305 310
Glu Trp Thr Gly Lys Asn Phe Val Pro Pro Leu Lys Thr
Leu Tyr Thr
315 320 325
Val Gln Asp Thr Tyr Asn Tyr Asn Asp Pro Thr Cys Gly
Asp Met Thr
330 335 340
Tyr Ile Cys Trp Pro Thr Val Ala Pro Ser Ser Ala Tyr
Val Tyr Lys
345 350 355
Gly Gly Lys Lys Ala Ile Ser Gly Trp Glu Asn Thr Leu
Leu Val Pro
360
365 370 375
Ser Leu Lys Arg Gly Val Ile Phe Arg Ile Lys Leu Asp
Pro Thr Tyr
380 385 390
Ser Thr Thr Tyr Asp Asp Ala Val Pro Met Phe Lys Ser
Asn Asn Arg
395 400 405
Tyr Arg Asp Val Ile Ala Ser Pro Asp Gly Asn Val Leu
Tyr Val Leu
410 415 420
Thr Asp Thr Ser Gly Asn Val Gln Lys Asp Asp Gly Ser
Val Thr Asn
425 430 435
Thr Leu Glu Asn Pro Gly Ser Leu Ile Arg Phe Thr Tyr
Lys Ala Gln
440
445 450 455
Ser
<210> 23
<211> 22
<212> DNA
<213> 人工的
<220>
<223>
PCR-引物
<400> 23
tagagttatt ttaccactcc ct
22
<210> 24
<211> 24
<212> DNA
<213> 人工的
<220>
<223>
PCR-引物
<400> 24
ggttaggtcc ctgatcacca atcg 24
<210> 25
<211> 22
<212> DNA
<213> 人工的
<220>
<223>
PCR-引物
<400> 25
cacaggtaca cctgccgcca ct
22
<210> 26
<211> 25
<212> DNA
<213> 人工的
<220>
<223>
PCR-引物
<400> 26
tacgaggacc caacaggaac tgagc 25
<210> 27
<211>
4508
<212> DNA
<213> 人工的
<220>
<223> 表达载体
<220>
<221> CDS
<222>
(139)..(720)
<220>
<221>
sig_肽
<222>
(139)..(213)
<220>
<221>
mat_肽
<222>
(214)..(720)
<400> 27
ccatcgaatg gccagatgat taattcctaa tttttgttga cactctatca
ttgatagagt 60
tattttacca ctccctatca gtgatagaga aaagtgaaat gaatagttcg
acaaaaatct 120
agataacgag ggcaaaaa atg aat aaa cat tta ttg gct aaa
att act tta 171
Met Asn Lys His Leu Leu Ala Lys Ile Thr Leu
-25 -20 -15
tta ggt gct gct cag cta ctt acg ctc aat tca gca ttt
gct gat gtc 219
Leu Gly Ala Ala Gln Leu Leu Thr Leu Asn Ser Ala Phe
Ala Asp Val
-10 -5 -1 1
cct ctt aca cca tct caa ttt gct aaa gcg aaa aca gaa
agc ttt gat 267
Pro Leu Thr Pro Ser Gln Phe Ala Lys Ala Lys Thr Glu
Ser Phe Asp
5 10 15
aag aaa gtt ctt cta tct aat tta aat aag cca cat gct
ttg ttg tgg 315
Lys Lys Val Leu Leu Ser Asn Leu Asn Lys Pro His Ala
Leu Leu Trp
20 25 30
gga ccg gat aat caa att tgg tta acg gag cgg gca aca
ggt aag att 363
Gly Pro Asp Asn Gln Ile Trp Leu Thr Glu Arg Ala Thr
Gly Lys Ile
35
40 45 50
cta aga gtt aat cca gag tcg ggc agt gta aaa aca gtt
ttt cag gtt 411
Leu Arg Val Asn Pro Glu Ser Gly Ser Val Lys Thr Val
Phe Gln Val
55 60 65
cct gag att gta aat gat gct gat gga caa aac ggt tta
ttg ggt ttt 459
Pro Glu Ile Val Asn Asp Ala Asp Gly Gln Asn Gly Leu
Leu Gly Phe
70 75 80
gcc ttt cat cct gac ttt aaa aat aat cct tat atc tat
gtt tca ggt 507
Ala Phe His Pro Asp Phe Lys Asn Asn Pro Tyr Ile Tyr
Val Ser Gly
85 90 95
aca ttt aaa aat ccg aaa tct aca gat aaa gaa tta ccg
aat caa act 555
Thr Phe Lys Asn Pro Lys Ser Thr Asp Lys Glu Leu Pro
Asn Gln Thr
100 105 110
att atc cgt cga tat acc tat aac aag gca aca gat acc
ctt gag aaa 603
Ile Ile Arg Arg Tyr Thr Tyr Asn Lys Ala Thr Asp Thr
Leu Glu Lys
115
120 125 130
cca gta gat tta ttg gca gga tta cct tca tcg aaa gac
cat cag tcg 651
Pro Val Asp Leu Leu Ala Gly Leu Pro Ser Ser Lys Asp
His Gln Ser
135 140 145
ggt cgt ctt gtg att ggt cca gac caa aag att tac tat
acg att ggt 699
Gly Arg Leu Val Ile Gly Pro Asp Gln Lys Ile Tyr Tyr
Thr Ile Gly
150 155 160
gat cag gga ccc aac agg aac tgagcggcaa agactatcat
acctatatgg 750
Asp Gln Gly Pro Asn Arg Asn
165
gtaaagtatt gcgcttaaat ctggatggaa gtattccaaa agataatcca
agctttaacg 810
gtgtaattag ccatatttat acgctcggtc atcgtaaccc acagggcttg
gcatttactc 870
caaatggtaa actgttgcaa tctgaacagg gtccaaactc tgatgatgaa
attaacctca 930
ttgtcaaagg tggtaactat ggctggccaa atgtagcggg ttataaagat
gatagtggtt 990
atgcctatgc aaattattcg gcagcaagca ataaagcaca aattaaagat
ttaggacaaa 1050
atggtttaaa agtggcggca ggtgtacctg tgatgaaaga gtctgaatgg
actggtaaaa 1110
actttgtacc gccgttaaaa actttatata ccgtccaaga tacctataac
tataatgacc 1170
caacttgtgg ggatatgacc tacatttgct ggccaacggt tgcgccgtca
tctgcttatg 1230
tctataaggg aggcaaaaaa gcaatttctg gttgggaaaa tacattattg
gttccatctt 1290
taaagcgcgg tgttattttc cgtattaagc tagatccaac ttacagtact
acttatgatg 1350
atgctgtgcc gatgtttaag agcaacaatc gttatcgtga cgtgattgca
agtccagatg 1410
ggaatgtttt atatgtattg actgatactt ccggaaatgt ccaaaaagat
gatggttctg 1470
taacgaatac attagaaaac ccaggatctc tgattagatt tacatataaa
gctcagagct 1530
gaaagcttga cctgtgaagt gaaaaatggc gcacattgtg cgacattttt
tttgtctgcc 1590
gtttaccgct actgcgtcac ggatctccac gcgccctgta gcggcgcatt
aagcgcggcg 1650
ggtgtggtgg ttacgcgcag cgtgaccgct acacttgcca gcgccctagc
gcccgctcct 1710
ttcgctttct tcccttcctt tctcgccacg ttcgccggct ttccccgtca
agctctaaat 1770
cgggggctcc ctttagggtt ccgatttagt gctttacggc acctcgaccc
caaaaaactt 1830
gattagggtg atggttcacg tagtgggcca tcgccctgat agacggtttt
tcgccctttg 1890
acgttggagt ccacgttctt taatagtgga ctcttgttcc aaactggaac
aacactcaac 1950
cctatctcgg tctattcttt tgatttataa gggattttgc cgatttcggc
ctattggtta 2010
aaaaatgagc tgatttaaca aaaatttaac gcgaatttta acaaaatatt
aacgcttaca 2070
atttcaggtg gcacttttcg gggaaatgtg cgcggaaccc ctatttgttt
atttttctaa 2130
atacattcaa atatgtatcc gctcatgaga caataaccct gataaatgct
tcaataatat 2190
tgaaaaagga agagtatgag tattcaacat ttccgtgtcg cccttattcc
cttttttgcg 2250
gcattttgcc ttcctgtttt tgctcaccca gaaacgctgg tgaaagtaaa
agatgctgaa 2310
gatcagttgg gtgcacgagt gggttacatc gaactggatc tcaacagcgg
taagatcctt 2370
gagagttttc gccccgaaga acgttttcca atgatgagca cttttaaagt
tctgctatgt 2430
ggcgcggtat tatcccgtat tgacgccggg caagagcaac tcggtcgccg
catacactat 2490
tctcagaatg acttggttga gtactcacca gtcacagaaa agcatcttac
ggatggcatg 2550
acagtaagag aattatgcag tgctgccata accatgagtg ataacactgc
ggccaactta 2610
cttctgacaa cgatcggagg accgaaggag ctaaccgctt ttttgcacaa
catgggggat 2670
catgtaactc gccttgatcg ttgggaaccg gagctgaatg aagccatacc
aaacgacgag 2730
cgtgacacca cgatgcctgt agcaatggca acaacgttgc gcaaactatt
aactggcgaa 2790
ctacttactc tagcttcccg gcaacaattg atagactgga tggaggcgga
taaagttgca 2850
ggaccacttc tgcgctcggc ccttccggct ggctggttta ttgctgataa
atctggagcc 2910
ggtgagcgtg gctctcgcgg tatcattgca gcactggggc cagatggtaa
gccctcccgt 2970
atcgtagtta tctacacgac ggggagtcag gcaactatgg atgaacgaaa
tagacagatc 3030
gctgagatag gtgcctcact gattaagcat tggtaggaat taatgatgtc
tcgtttagat 3090
aaaagtaaag tgattaacag cgcattagag ctgcttaatg aggtcggaat
cgaaggttta 3150
acaacccgta aactcgccca gaagctaggt gtagagcagc ctacattgta
ttggcatgta 3210
aaaaataagc gggctttgct cgacgcctta gccattgaga tgttagatag
gcaccatact 3270
cacttttgcc ctttagaagg ggaaagctgg caagattttt tacgtaataa
cgctaaaagt 3330
tttagatgtg ctttactaag tcatcgcgat ggagcaaaag tacatttagg
tacacggcct 3390
acagaaaaac agtatgaaac tctcgaaaat caattagcct ttttatgcca
acaaggtttt 3450
tcactagaga atgcattata tgcactcagc gcagtggggc attttacttt
aggttgcgta 3510
ttggaagatc aagagcatca agtcgctaaa gaagaaaggg aaacacctac
tactgatagt 3570
atgccgccat tattacgaca agctatcgaa ttatttgatc accaaggtgc
agagccagcc 3630
ttcttattcg gccttgaatt gatcatatgc ggattagaaa aacaacttaa
atgtgaaagt 3690
gggtcttaaa agcagcataa cctttttccg tgatggtaac ttcactagtt
taaaaggatc 3750
taggtgaaga tcctttttga taatctcatg accaaaatcc cttaacgtga
gttttcgttc 3810
cactgagcgt cagaccccgt agaaaagatc aaaggatctt cttgagatcc
tttttttctg 3870
cgcgtaatct gctgcttgca aacaaaaaaa ccaccgctac cagcggtggt
ttgtttgccg 3930
gatcaagagc taccaactct ttttccgaag gtaactggct tcagcagagc
gcagatacca 3990
aatactgtcc ttctagtgta gccgtagtta ggccaccact tcaagaactc
tgtagcaccg 4050
cctacatacc tcgctctgct aatcctgtta ccagtggctg ctgccagtgg
cgataagtcg 4110
tgtcttaccg ggttggactc aagacgatag ttaccggata aggcgcagcg
gtcgggctga 4170
acggggggtt cgtgcacaca gcccagcttg gagcgaacga cctacaccga
actgagatac 4230
ctacagcgtg agctatgaga aagcgccacg cttcccgaag ggagaaaggc
ggacaggtat 4290
ccggtaagcg gcagggtcgg aacaggagag cgcacgaggg agcttccagg
gggaaacgcc 4350
tggtatcttt atagtcctgt cgggtttcgc cacctctgac ttgagcgtcg
atttttgtga 4410
tgctcgtcag gggggcggag cctatggaaa aacgccagca acgcggcctt
tttacggttc 4470
ctggcctttt gctggccttt tgctcacatg acccgaca 4508
<210> 28
<211> 194
<212> PRT
<213> 人工的
<220>
<223> 合成构建体
<400> 28
Met Asn Lys His Leu Leu Ala Lys Ile Thr Leu Leu Gly
Ala Ala Gln
-25
-20 -15 -10
Leu Leu Thr Leu Asn Ser Ala Phe Ala Asp Val Pro Leu
Thr Pro Ser
-5 -1
1 5
Gln Phe Ala Lys Ala Lys Thr Glu Ser Phe Asp Lys Lys
Val Leu Leu
10 15 20
Ser Asn Leu Asn Lys Pro His Ala Leu Leu Trp Gly Pro
Asp Asn Gln
25 30 35
Ile Trp Leu Thr Glu Arg Ala Thr Gly Lys Ile Leu Arg
Val Asn Pro
40
45 50 55
Glu Ser Gly Ser Val Lys Thr Val Phe Gln Val Pro Glu
Ile Val Asn
60 65 70
Asp Ala Asp Gly Gln Asn Gly Leu Leu Gly Phe Ala Phe
His Pro Asp
75 80 85
Phe Lys Asn Asn Pro Tyr Ile Tyr Val Ser Gly Thr Phe
Lys Asn Pro
90 95 100
Lys Ser Thr Asp Lys Glu Leu Pro Asn Gln Thr Ile Ile
Arg Arg Tyr
105 110 115
Thr Tyr Asn Lys Ala Thr Asp Thr Leu Glu Lys Pro Val
Asp Leu Leu
120
125 130 135
Ala Gly Leu Pro Ser Ser Lys Asp His Gln Ser Gly Arg
Leu Val Ile
140 145 150
Gly Pro Asp Gln Lys Ile Tyr Tyr Thr Ile Gly Asp Gln
Gly Pro Asn
155 160 165
Arg Asn
<210> 29
<211> 36
<212> DNA
<213> 人工的
<220>
<223>
PCR-引物
<220>
<221> 多样性特征
<222>
(13)..(24)
<223> n 对应于
a, c, g 或 t; k 对应于 g 或 t
<400> 29
cgacgtaacc agnnknnknn knnkctggct tacctg 36
<210> 30
<211> 38
<212> DNA
<213> 人工的
<220>
<223>
PCR-引物
<400> 30
gtgtgctgtg cctggttcgg caggaacagg taagccag 38
<210> 31
<211> 29
<212> DNA
<213> 人工的
<220>
<223>
PCR-引物
<400> 31
cctacctacg actcttccga cgtaaccag 29
<210> 32
<211> 30
<212> DNA
<213> 人工的
<220>
<223>
PCR-引物
<400> 32
ccatgctctt cagtcggagt gtgctgtgcc 30
<210> 33
<211> 64
<212> DNA
<213> 人工的
<220>
<223> 具有四个随机位置的插入片段
<220>
<221> 多样性特征
<222>
(12)..(23)
<223> n 为 a,
c, g 或 t
<400> 33
gacgtaacca gnnnnnnnnn nnnctggctt acctgttcct gccgaaccag
gcacagcaca 60
ctcc
64
<210> 34
<211> 22
<212> DNA
<213> 人工的
<220>
<223> 测序引物
<400> 34
caccgttaaa gcttggatta tc 22
<210> 35
<211> 21
<212> DNA
<213> 人工的
<220>
<223> 测序引物
<400> 35
ccttcatcga aagaccatca g
21
Claims (13)
1.可溶性吡咯并喹啉醌依赖性葡萄糖脱氢酶(sPQQGDH),其特征在于,与不动杆菌属的一种野生型菌株诸如,例如,醋酸钙不动杆菌(Acinetobacter
calcoaceticus) LMD 79.41的sPQQGDH相比,其在底物结合区域的范围内具有3-5个氨基酸的插入。
2.如权利要求1所述的sPQQGDH,其特征在于,插入位点距离底物结合区域不超过5埃。
3.如权利要求1或2所述的sPQQGDH,其特征在于,基于野生型醋酸钙不动杆菌LMD 79.41的序列,插入位点在位置Q168和L169之间。
4.如权利要求1-3中任一项所述的sPQQGDH,其特征在于,其通过诱变由PT16、KOZ62、KOZ65、PTN69、KG106、PTN26、PT15、KGN80、KG140、KGN34、KGN25、KGN100系列的菌株产生。
5.如权利要求1-4中任一项所述的sPQQGDH,其具有3个氨基酸的插入,其中插入的氨基酸在位置1具有A或G,在位置2具有Y、F或W,和在位置3具有Q、L、V或I。
6.如权利要求1-4中任一项所述的sPQQGDH,其具有4个氨基酸的插入,其中插入的氨基酸在位置1具有A或G,在位置2具有G、D或L,在位置3具有R或A,和在位置4具有M或V。
7.如权利要求1-4中任一项所述的sPQQGDH,其具有5个氨基酸的插入,其中插入的氨基酸在位置1具有A、M、S或V,在位置2具有E、G或S,在位置3具有H、K、R、S或T,在位置4具有F、H、I、L、N、V或Y,和在位置5具有F、H、L、N、Q、T或Y。
8.如权利要求1-4中任一项所述的sPQQGDH,其具有选自系列SEQ ID 1-SEQ ID 18的插入序列。
9.基因,其编码一种如权利要求1-8中任一项所述的蛋白质。
10.载体,其特征在于,其在插入位点的范围内具有一个唯一的限制位点,以便可以在该位点插入编码如权利要求1-6中任一项所述的sPQQGDH的寡核苷酸序列。
11.如权利要求10所述的载体,其具有序列SEQ ID 27。
12.用于生产和鉴别如权利要求1-8中任一项所述的sPQQGDH的方法,其特征在于,在第一步中生产在插入位点的范围内具有唯一的限制位点的载体,在第二步中将多种不同的寡核苷酸序列插入载体中,在第三步中将这样获得的重组载体转化到宿主细菌中并使细菌增殖,在第四步中筛选细菌产生的sPQQGDH,以便测定sPQQGDH对至少两种不同的糖的单独活性以及至少一种糖与至少另一种糖的干扰作用,和在第五步中选择与未修饰的野生菌株的sPQQGDH相比具有改善的底物特异性的那些sPQQGDH。
13.葡萄糖传感器,其包含权利要求1-8的sPQQGDH。
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| DE102008030435A DE102008030435A1 (de) | 2008-06-26 | 2008-06-26 | Neuartige Varianten PQQ-abhängiger Glukosehydrogenase mit verbesserter Substratspezifität |
| DE102008030435.2 | 2008-06-26 | ||
| PCT/EP2009/004350 WO2009156083A1 (de) | 2008-06-26 | 2009-06-17 | Neuartige varianten pqq-abhängiger glukosedehydrogenase mit verbesserter substratspezifität |
Related Child Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| CN201310472347.1A Division CN103540572A (zh) | 2008-06-26 | 2009-06-17 | 具有改善的底物特异性的pqq-依赖性葡萄糖脱氢酶的新变体 |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| CN102076846A true CN102076846A (zh) | 2011-05-25 |
Family
ID=40972803
Family Applications (2)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| CN200980124063.5A Pending CN102076846A (zh) | 2008-06-26 | 2009-06-17 | 具有改善的底物特异性的pqq-依赖性葡萄糖脱氢酶的新变体 |
| CN201310472347.1A Pending CN103540572A (zh) | 2008-06-26 | 2009-06-17 | 具有改善的底物特异性的pqq-依赖性葡萄糖脱氢酶的新变体 |
Family Applications After (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| CN201310472347.1A Pending CN103540572A (zh) | 2008-06-26 | 2009-06-17 | 具有改善的底物特异性的pqq-依赖性葡萄糖脱氢酶的新变体 |
Country Status (6)
| Country | Link |
|---|---|
| US (1) | US8580547B2 (zh) |
| EP (1) | EP2294188A1 (zh) |
| JP (1) | JP2011525361A (zh) |
| CN (2) | CN102076846A (zh) |
| DE (1) | DE102008030435A1 (zh) |
| WO (1) | WO2009156083A1 (zh) |
Families Citing this family (4)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| EP2465936A1 (en) | 2010-12-20 | 2012-06-20 | LEK Pharmaceuticals d.d. | Enzymatic synthesis of statins and intermediates thereof |
| US20130337485A1 (en) | 2010-12-20 | 2013-12-19 | Lek Pharmaceuticals D.D | Enzymatic synthesis of active pharmaceutical ingredient and intermediates thereof |
| CN111073866B (zh) | 2015-10-29 | 2024-03-19 | 英科隆生物技术(杭州)有限公司 | PQQ-sGDH突变体、聚核苷酸及其在葡萄糖检测中的应用 |
| CN109085269B (zh) * | 2018-08-15 | 2021-03-09 | 江苏省原子医学研究所 | PQQ与Lys及Arg反应行为研究的方法 |
Family Cites Families (9)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| ATE313790T1 (de) | 1999-10-05 | 2006-01-15 | Matsushita Electric Ind Co Ltd | Glukosesensor |
| CA2427029C (en) | 2000-10-27 | 2010-02-02 | F. Hoffmann-La Roche Ag | Variants of soluble pyrroloquinoline quinone-dependent glucose dehydrogenase |
| US7476525B2 (en) | 2002-05-27 | 2009-01-13 | Toyo Boseki Kabushiki Kaisha | Modified pyrroloquinoline quinone (PQQ) dependent glucose dehydrogenase with superior substrate specificity and stability |
| DE10320259A1 (de) | 2003-05-07 | 2004-11-25 | Bayer Technology Services Gmbh | Neuartige Glukose Dehydrogenase und ihre Herstellung |
| EP1666586B1 (en) | 2003-09-08 | 2009-10-21 | Toyo Boseki Kabushiki Kaisha | Pyrroloquinoline quinone (pqq)-dependent glucose dehydrogenase modification having excellent substrate specificity |
| JP2007043983A (ja) * | 2005-08-11 | 2007-02-22 | Toyobo Co Ltd | 基質特異性に優れた改変型ピロロキノリンキノン依存性グルコースデヒドロゲナーゼ |
| WO2006085509A1 (ja) * | 2005-02-08 | 2006-08-17 | Toyo Boseki Kabushiki Kaisha | 基質特異性に優れた改変型ピロロキノリンキノン依存性グルコースデヒドロゲナーゼ |
| JP2007000020A (ja) * | 2005-06-21 | 2007-01-11 | Toyobo Co Ltd | 基質特異性に優れたピロロキノリンキノン(pqq)依存性グルコースデヒドロゲナーゼ改変体 |
| JP2006217810A (ja) * | 2005-02-08 | 2006-08-24 | Toyobo Co Ltd | 基質特異性に優れたピロロキノリンキノン(pqq)依存性グルコースデヒドロゲナーゼ改変体 |
-
2008
- 2008-06-26 DE DE102008030435A patent/DE102008030435A1/de not_active Withdrawn
-
2009
- 2009-06-17 US US12/999,465 patent/US8580547B2/en not_active Expired - Fee Related
- 2009-06-17 WO PCT/EP2009/004350 patent/WO2009156083A1/de active Application Filing
- 2009-06-17 JP JP2011515164A patent/JP2011525361A/ja active Pending
- 2009-06-17 CN CN200980124063.5A patent/CN102076846A/zh active Pending
- 2009-06-17 EP EP09768932A patent/EP2294188A1/de not_active Withdrawn
- 2009-06-17 CN CN201310472347.1A patent/CN103540572A/zh active Pending
Also Published As
| Publication number | Publication date |
|---|---|
| DE102008030435A1 (de) | 2010-01-07 |
| US20110236920A1 (en) | 2011-09-29 |
| WO2009156083A1 (de) | 2009-12-30 |
| US8580547B2 (en) | 2013-11-12 |
| EP2294188A1 (de) | 2011-03-16 |
| JP2011525361A (ja) | 2011-09-22 |
| CN103540572A (zh) | 2014-01-29 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| AU2002215966B2 (en) | Variants of soluble pyrroloquinoline quinone-dependent glucose dehydrogenase | |
| EP2010649B1 (en) | Improved mutants of pyrroloquinoline quinone dependent soluble glucose dehydrogenase | |
| EP1167519A1 (en) | Glucose dehydrogenase | |
| CN101040044B (zh) | 吡咯并喹啉醌依赖性葡糖脱氢酶的热稳定突变体 | |
| CN100549167C (zh) | 包含氨基酸插入的遗传改造的吡咯并喹啉醌依赖性葡萄糖脱氢酶 | |
| JP2010057427A (ja) | グルコース脱水素酵素およびグルコースの電気化学測定法 | |
| CN102076846A (zh) | 具有改善的底物特异性的pqq-依赖性葡萄糖脱氢酶的新变体 | |
| KR102159807B1 (ko) | 디히드로게나아제 활성이 향상된 아마도리아제 | |
| JP6635913B2 (ja) | 比活性が向上したフラビン結合型グルコースデヒドロゲナーゼ | |
| JP6101011B2 (ja) | フラビン結合型グルコースデヒドロゲナーゼを含む組成物の安定性を向上する方法 | |
| JP6934720B2 (ja) | 基質特異性が向上したフラビン結合型グルコースデヒドロゲナーゼ | |
| JPWO2017183717A1 (ja) | HbA1cデヒドロゲナーゼ | |
| JP2011050300A (ja) | グルコースセンサ | |
| JP5622321B2 (ja) | 耐熱性1,5−アンヒドログルシトール脱水素酵素及びそれを用いた1,5−アンヒドログルシトールの測定法 | |
| JP5899616B2 (ja) | フラビンアデニンジヌクレオチド依存性グルコースデヒドロゲナーゼの温度依存性を改善する方法 | |
| JP2012029677A (ja) | フラビンアデニンジヌクレオチド依存性グルコースデヒドロゲナーゼの基質特異性を改善するための方法 | |
| AU2006201542A1 (en) | Variants of soluble pyrroloquinoline quinone-dependent glucose dehydrogenase |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| C06 | Publication | ||
| PB01 | Publication | ||
| C10 | Entry into substantive examination | ||
| SE01 | Entry into force of request for substantive examination | ||
| ASS | Succession or assignment of patent right |
Owner name: BAYER INTELLECTUAL PROPERTY GMBH Free format text: FORMER OWNER: BAYER AG Effective date: 20130807 |
|
| C41 | Transfer of patent application or patent right or utility model | ||
| TA01 | Transfer of patent application right |
Effective date of registration: 20130807 Address after: German Monheim Applicant after: Bayer Pharma Aktiengesellschaft Address before: Germany Leverkusen Applicant before: Bayer Ag |
|
| C02 | Deemed withdrawal of patent application after publication (patent law 2001) | ||
| WD01 | Invention patent application deemed withdrawn after publication |
Application publication date: 20110525 |