BR112013000552B1 - estratégias de sequenciamento de região de interesse genômica em 3d - Google Patents
estratégias de sequenciamento de região de interesse genômica em 3d Download PDFInfo
- Publication number
- BR112013000552B1 BR112013000552B1 BR112013000552-1A BR112013000552A BR112013000552B1 BR 112013000552 B1 BR112013000552 B1 BR 112013000552B1 BR 112013000552 A BR112013000552 A BR 112013000552A BR 112013000552 B1 BR112013000552 B1 BR 112013000552B1
- Authority
- BR
- Brazil
- Prior art keywords
- dna
- fragments
- linked
- interest
- sequence
- Prior art date
Links
- 238000011451 sequencing strategy Methods 0.000 title 1
- 108020004414 DNA Proteins 0.000 claims abstract description 385
- 239000012634 fragment Substances 0.000 claims abstract description 352
- 125000003729 nucleotide group Chemical group 0.000 claims abstract description 124
- 239000002773 nucleotide Substances 0.000 claims abstract description 121
- 238000000034 method Methods 0.000 claims abstract description 104
- 238000004132 cross linking Methods 0.000 claims abstract description 38
- 239000000523 sample Substances 0.000 claims description 78
- 230000035772 mutation Effects 0.000 claims description 77
- 239000013615 primer Substances 0.000 claims description 65
- 238000013467 fragmentation Methods 0.000 claims description 54
- 238000006062 fragmentation reaction Methods 0.000 claims description 54
- 108091008146 restriction endonucleases Proteins 0.000 claims description 43
- 108091028043 Nucleic acid sequence Proteins 0.000 claims description 27
- 230000027455 binding Effects 0.000 claims description 20
- 238000012165 high-throughput sequencing Methods 0.000 claims description 19
- FGUUSXIOTUKUDN-IBGZPJMESA-N C1(=CC=CC=C1)N1C2=C(NC([C@H](C1)NC=1OC(=NN=1)C1=CC=CC=C1)=O)C=CC=C2 Chemical compound C1(=CC=CC=C1)N1C2=C(NC([C@H](C1)NC=1OC(=NN=1)C1=CC=CC=C1)=O)C=CC=C2 FGUUSXIOTUKUDN-IBGZPJMESA-N 0.000 claims description 12
- 238000000527 sonication Methods 0.000 claims description 11
- 238000003780 insertion Methods 0.000 claims description 10
- 230000037431 insertion Effects 0.000 claims description 10
- 238000012217 deletion Methods 0.000 claims description 9
- GNFTZDOKVXKIBK-UHFFFAOYSA-N 3-(2-methoxyethoxy)benzohydrazide Chemical compound COCCOC1=CC=CC(C(=O)NN)=C1 GNFTZDOKVXKIBK-UHFFFAOYSA-N 0.000 claims description 8
- 230000037430 deletion Effects 0.000 claims description 8
- 230000002255 enzymatic effect Effects 0.000 claims description 7
- 230000008439 repair process Effects 0.000 claims description 6
- 230000005945 translocation Effects 0.000 claims description 6
- 238000000605 extraction Methods 0.000 claims description 5
- 239000003999 initiator Substances 0.000 claims description 5
- 239000003155 DNA primer Substances 0.000 claims description 4
- 230000008878 coupling Effects 0.000 claims description 4
- 238000010168 coupling process Methods 0.000 claims description 4
- 238000005859 coupling reaction Methods 0.000 claims description 4
- 238000001502 gel electrophoresis Methods 0.000 claims description 4
- 238000004587 chromatography analysis Methods 0.000 claims description 3
- 230000002759 chromosomal effect Effects 0.000 claims description 3
- 239000013074 reference sample Substances 0.000 claims description 3
- YTAHJIFKAKIKAV-XNMGPUDCSA-N [(1R)-3-morpholin-4-yl-1-phenylpropyl] N-[(3S)-2-oxo-5-phenyl-1,3-dihydro-1,4-benzodiazepin-3-yl]carbamate Chemical compound O=C1[C@H](N=C(C2=C(N1)C=CC=C2)C1=CC=CC=C1)NC(O[C@H](CCN1CCOCC1)C1=CC=CC=C1)=O YTAHJIFKAKIKAV-XNMGPUDCSA-N 0.000 claims description 2
- 238000010276 construction Methods 0.000 claims description 2
- 238000000432 density-gradient centrifugation Methods 0.000 claims description 2
- 238000012163 sequencing technique Methods 0.000 abstract description 38
- 210000004027 cell Anatomy 0.000 description 46
- WSFSSNUMVMOOMR-UHFFFAOYSA-N Formaldehyde Chemical compound O=C WSFSSNUMVMOOMR-UHFFFAOYSA-N 0.000 description 30
- 238000003752 polymerase chain reaction Methods 0.000 description 27
- 230000003321 amplification Effects 0.000 description 21
- 238000003199 nucleic acid amplification method Methods 0.000 description 21
- 108090000623 proteins and genes Proteins 0.000 description 21
- 108700028369 Alleles Proteins 0.000 description 18
- 239000000047 product Substances 0.000 description 17
- 101150072950 BRCA1 gene Proteins 0.000 description 13
- 210000000349 chromosome Anatomy 0.000 description 13
- 102000004190 Enzymes Human genes 0.000 description 12
- 108090000790 Enzymes Proteins 0.000 description 12
- 102000004169 proteins and genes Human genes 0.000 description 12
- 238000006243 chemical reaction Methods 0.000 description 11
- 108700040618 BRCA1 Genes Proteins 0.000 description 10
- 102000053602 DNA Human genes 0.000 description 10
- 108091034117 Oligonucleotide Proteins 0.000 description 9
- 230000000875 corresponding effect Effects 0.000 description 9
- 238000005516 engineering process Methods 0.000 description 9
- 230000003993 interaction Effects 0.000 description 9
- 210000001519 tissue Anatomy 0.000 description 9
- ISAKRJDGNUQOIC-UHFFFAOYSA-N Uracil Chemical compound O=C1C=CNC(=O)N1 ISAKRJDGNUQOIC-UHFFFAOYSA-N 0.000 description 8
- 239000004971 Cross linker Substances 0.000 description 7
- 206010028980 Neoplasm Diseases 0.000 description 7
- 150000007523 nucleic acids Chemical class 0.000 description 7
- 238000011282 treatment Methods 0.000 description 7
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N (+)-Biotin Chemical compound N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 description 6
- UYTPUPDQBNUYGX-UHFFFAOYSA-N guanine Chemical compound O=C1NC(N)=NC2=C1N=CN2 UYTPUPDQBNUYGX-UHFFFAOYSA-N 0.000 description 6
- 102000039446 nucleic acids Human genes 0.000 description 6
- 108020004707 nucleic acids Proteins 0.000 description 6
- 239000007787 solid Substances 0.000 description 6
- LSNNMFCWUKXFEE-UHFFFAOYSA-M Bisulfite Chemical compound OS([O-])=O LSNNMFCWUKXFEE-UHFFFAOYSA-M 0.000 description 5
- 238000013459 approach Methods 0.000 description 5
- 201000011510 cancer Diseases 0.000 description 5
- 238000009396 hybridization Methods 0.000 description 5
- 238000000926 separation method Methods 0.000 description 5
- 238000001574 biopsy Methods 0.000 description 4
- 230000000295 complement effect Effects 0.000 description 4
- OPTASPLRGRRNAP-UHFFFAOYSA-N cytosine Chemical compound NC=1C=CNC(=O)N=1 OPTASPLRGRRNAP-UHFFFAOYSA-N 0.000 description 4
- 230000029087 digestion Effects 0.000 description 4
- 201000010099 disease Diseases 0.000 description 4
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 4
- 238000012545 processing Methods 0.000 description 4
- 239000000126 substance Substances 0.000 description 4
- RWQNBRDOKXIBIV-UHFFFAOYSA-N thymine Chemical compound CC1=CNC(=O)NC1=O RWQNBRDOKXIBIV-UHFFFAOYSA-N 0.000 description 4
- 229940035893 uracil Drugs 0.000 description 4
- 229930024421 Adenine Natural products 0.000 description 3
- GFFGJBXGBJISGV-UHFFFAOYSA-N Adenine Chemical compound NC1=NC=NC2=C1N=CN2 GFFGJBXGBJISGV-UHFFFAOYSA-N 0.000 description 3
- 108091093088 Amplicon Proteins 0.000 description 3
- 102000036365 BRCA1 Human genes 0.000 description 3
- 108700020463 BRCA1 Proteins 0.000 description 3
- 230000000970 DNA cross-linking effect Effects 0.000 description 3
- 238000012408 PCR amplification Methods 0.000 description 3
- 108091005804 Peptidases Proteins 0.000 description 3
- 239000004365 Protease Substances 0.000 description 3
- 102100037486 Reverse transcriptase/ribonuclease H Human genes 0.000 description 3
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 description 3
- 229960000643 adenine Drugs 0.000 description 3
- 238000004458 analytical method Methods 0.000 description 3
- 238000003556 assay Methods 0.000 description 3
- 229960002685 biotin Drugs 0.000 description 3
- 235000020958 biotin Nutrition 0.000 description 3
- 239000011616 biotin Substances 0.000 description 3
- 238000004113 cell culture Methods 0.000 description 3
- 238000004590 computer program Methods 0.000 description 3
- 210000001840 diploid cell Anatomy 0.000 description 3
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 2
- KDCGOANMDULRCW-UHFFFAOYSA-N 7H-purine Chemical compound N1=CNC2=NC=NC2=C1 KDCGOANMDULRCW-UHFFFAOYSA-N 0.000 description 2
- 241000894006 Bacteria Species 0.000 description 2
- 206010006187 Breast cancer Diseases 0.000 description 2
- 208000026310 Breast neoplasm Diseases 0.000 description 2
- 108010077544 Chromatin Proteins 0.000 description 2
- 102000012410 DNA Ligases Human genes 0.000 description 2
- 108010061982 DNA Ligases Proteins 0.000 description 2
- 238000001712 DNA sequencing Methods 0.000 description 2
- 230000006820 DNA synthesis Effects 0.000 description 2
- 108060002716 Exonuclease Proteins 0.000 description 2
- 108020002230 Pancreatic Ribonuclease Proteins 0.000 description 2
- 102000005891 Pancreatic ribonuclease Human genes 0.000 description 2
- 238000012300 Sequence Analysis Methods 0.000 description 2
- 241000700605 Viruses Species 0.000 description 2
- YTRQFSDWAXHJCC-UHFFFAOYSA-N chloroform;phenol Chemical compound ClC(Cl)Cl.OC1=CC=CC=C1 YTRQFSDWAXHJCC-UHFFFAOYSA-N 0.000 description 2
- 210000003483 chromatin Anatomy 0.000 description 2
- 238000007796 conventional method Methods 0.000 description 2
- 238000013461 design Methods 0.000 description 2
- 238000010790 dilution Methods 0.000 description 2
- 239000012895 dilution Substances 0.000 description 2
- 230000000694 effects Effects 0.000 description 2
- 102000013165 exonuclease Human genes 0.000 description 2
- 238000011049 filling Methods 0.000 description 2
- 230000007614 genetic variation Effects 0.000 description 2
- 239000003550 marker Substances 0.000 description 2
- 239000000463 material Substances 0.000 description 2
- WSFSSNUMVMOOMR-NJFSPNSNSA-N methanone Chemical compound O=[14CH2] WSFSSNUMVMOOMR-NJFSPNSNSA-N 0.000 description 2
- 230000011987 methylation Effects 0.000 description 2
- 238000007069 methylation reaction Methods 0.000 description 2
- 239000000203 mixture Substances 0.000 description 2
- 238000012986 modification Methods 0.000 description 2
- 230000004048 modification Effects 0.000 description 2
- 239000008188 pellet Substances 0.000 description 2
- 229920000642 polymer Polymers 0.000 description 2
- 102000040430 polynucleotide Human genes 0.000 description 2
- 108091033319 polynucleotide Proteins 0.000 description 2
- 239000002157 polynucleotide Substances 0.000 description 2
- 230000008569 process Effects 0.000 description 2
- 238000000746 purification Methods 0.000 description 2
- 238000003908 quality control method Methods 0.000 description 2
- 238000010008 shearing Methods 0.000 description 2
- 239000000243 solution Substances 0.000 description 2
- 239000006228 supernatant Substances 0.000 description 2
- 238000002560 therapeutic procedure Methods 0.000 description 2
- 229940113082 thymine Drugs 0.000 description 2
- LRSASMSXMSNRBT-UHFFFAOYSA-N 5-methylcytosine Chemical group CC1=CNC(=O)N=C1N LRSASMSXMSNRBT-UHFFFAOYSA-N 0.000 description 1
- 108700010154 BRCA2 Genes Proteins 0.000 description 1
- 208000035143 Bacterial infection Diseases 0.000 description 1
- 230000004544 DNA amplification Effects 0.000 description 1
- 230000009946 DNA mutation Effects 0.000 description 1
- 230000004568 DNA-binding Effects 0.000 description 1
- 102000016928 DNA-directed DNA polymerase Human genes 0.000 description 1
- 108010014303 DNA-directed DNA polymerase Proteins 0.000 description 1
- 206010061818 Disease progression Diseases 0.000 description 1
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 1
- 241000196324 Embryophyta Species 0.000 description 1
- 240000007108 Fuchsia magellanica Species 0.000 description 1
- 241000233866 Fungi Species 0.000 description 1
- 108091027305 Heteroduplex Proteins 0.000 description 1
- 108010033040 Histones Proteins 0.000 description 1
- 102000006947 Histones Human genes 0.000 description 1
- 241000282412 Homo Species 0.000 description 1
- 101000836075 Homo sapiens Serpin B9 Proteins 0.000 description 1
- 101000661807 Homo sapiens Suppressor of tumorigenicity 14 protein Proteins 0.000 description 1
- 102000003960 Ligases Human genes 0.000 description 1
- 108090000364 Ligases Proteins 0.000 description 1
- 241001465754 Metazoa Species 0.000 description 1
- 102100030569 Nuclear receptor corepressor 2 Human genes 0.000 description 1
- 101710153660 Nuclear receptor corepressor 2 Proteins 0.000 description 1
- 229910019142 PO4 Inorganic materials 0.000 description 1
- 108091093037 Peptide nucleic acid Proteins 0.000 description 1
- CZPWVGJYEJSRLH-UHFFFAOYSA-N Pyrimidine Chemical compound C1=CN=CN=C1 CZPWVGJYEJSRLH-UHFFFAOYSA-N 0.000 description 1
- 108020004511 Recombinant DNA Proteins 0.000 description 1
- 108091028664 Ribonucleotide Proteins 0.000 description 1
- 239000006146 Roswell Park Memorial Institute medium Substances 0.000 description 1
- 102100025517 Serpin B9 Human genes 0.000 description 1
- 108010090804 Streptavidin Proteins 0.000 description 1
- 101100439974 Streptococcus pneumoniae serotype 4 (strain ATCC BAA-334 / TIGR4) clpE gene Proteins 0.000 description 1
- 208000036142 Viral infection Diseases 0.000 description 1
- 230000001464 adherent effect Effects 0.000 description 1
- 208000022362 bacterial infectious disease Diseases 0.000 description 1
- 239000000872 buffer Substances 0.000 description 1
- 210000003855 cell nucleus Anatomy 0.000 description 1
- 239000003795 chemical substances by application Substances 0.000 description 1
- 238000003776 cleavage reaction Methods 0.000 description 1
- 230000002596 correlated effect Effects 0.000 description 1
- 239000003431 cross linking reagent Substances 0.000 description 1
- 210000004748 cultured cell Anatomy 0.000 description 1
- 238000004163 cytometry Methods 0.000 description 1
- 229940104302 cytosine Drugs 0.000 description 1
- 239000005547 deoxyribonucleotide Substances 0.000 description 1
- 125000002637 deoxyribonucleotide group Chemical group 0.000 description 1
- 238000001514 detection method Methods 0.000 description 1
- 238000011161 development Methods 0.000 description 1
- 238000003745 diagnosis Methods 0.000 description 1
- 229940042399 direct acting antivirals protease inhibitors Drugs 0.000 description 1
- 230000005750 disease progression Effects 0.000 description 1
- 238000009826 distribution Methods 0.000 description 1
- 238000006911 enzymatic reaction Methods 0.000 description 1
- 230000007717 exclusion Effects 0.000 description 1
- 230000002349 favourable effect Effects 0.000 description 1
- 238000000855 fermentation Methods 0.000 description 1
- 230000004151 fermentation Effects 0.000 description 1
- -1 for example Proteins 0.000 description 1
- 238000010438 heat treatment Methods 0.000 description 1
- 210000005260 human cell Anatomy 0.000 description 1
- 125000002887 hydroxy group Chemical group [H]O* 0.000 description 1
- 238000001727 in vivo Methods 0.000 description 1
- 238000011835 investigation Methods 0.000 description 1
- 238000005304 joining Methods 0.000 description 1
- 239000012139 lysis buffer Substances 0.000 description 1
- 230000036210 malignancy Effects 0.000 description 1
- 238000002493 microarray Methods 0.000 description 1
- 244000005700 microbiome Species 0.000 description 1
- 239000004005 microsphere Substances 0.000 description 1
- 238000002156 mixing Methods 0.000 description 1
- 238000010369 molecular cloning Methods 0.000 description 1
- 230000007935 neutral effect Effects 0.000 description 1
- 238000007481 next generation sequencing Methods 0.000 description 1
- 210000004940 nucleus Anatomy 0.000 description 1
- 238000011275 oncology therapy Methods 0.000 description 1
- 238000005457 optimization Methods 0.000 description 1
- 230000008520 organization Effects 0.000 description 1
- 239000000137 peptide hydrolase inhibitor Substances 0.000 description 1
- 230000000737 periodic effect Effects 0.000 description 1
- NBIIXXVUZAFLBC-UHFFFAOYSA-K phosphate Chemical compound [O-]P([O-])([O-])=O NBIIXXVUZAFLBC-UHFFFAOYSA-K 0.000 description 1
- 239000010452 phosphate Substances 0.000 description 1
- 238000002360 preparation method Methods 0.000 description 1
- 238000004393 prognosis Methods 0.000 description 1
- 239000002994 raw material Substances 0.000 description 1
- 239000002336 ribonucleotide Substances 0.000 description 1
- 125000002652 ribonucleotide group Chemical group 0.000 description 1
- 238000005096 rolling process Methods 0.000 description 1
- 238000007480 sanger sequencing Methods 0.000 description 1
- 230000007017 scission Effects 0.000 description 1
- 238000012216 screening Methods 0.000 description 1
- 238000005728 strengthening Methods 0.000 description 1
- 239000000758 substrate Substances 0.000 description 1
- 238000009281 ultraviolet germicidal irradiation Methods 0.000 description 1
- 238000010200 validation analysis Methods 0.000 description 1
- 230000009385 viral infection Effects 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6869—Methods for sequencing
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q1/00—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions
- C12Q1/68—Measuring or testing processes involving enzymes, nucleic acids or microorganisms; Compositions therefor; Processes of preparing such compositions involving nucleic acids
- C12Q1/6844—Nucleic acid amplification reactions
- C12Q1/6853—Nucleic acid amplification reactions using modified primers or templates
- C12Q1/6855—Ligating adaptors
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2521/00—Reaction characterised by the enzymatic activity
- C12Q2521/30—Phosphoric diester hydrolysing, i.e. nuclease
- C12Q2521/301—Endonuclease
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2521/00—Reaction characterised by the enzymatic activity
- C12Q2521/50—Other enzymatic activities
- C12Q2521/501—Ligase
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2523/00—Reactions characterised by treatment of reaction samples
- C12Q2523/10—Characterised by chemical treatment
- C12Q2523/101—Crosslinking agents, e.g. psoralen
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2523/00—Reactions characterised by treatment of reaction samples
- C12Q2523/10—Characterised by chemical treatment
- C12Q2523/107—Chemical cleaving agents
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2525/00—Reactions involving modified oligonucleotides, nucleic acids, or nucleotides
- C12Q2525/30—Oligonucleotides characterised by their secondary structure
- C12Q2525/307—Circular oligonucleotides
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Q—MEASURING OR TESTING PROCESSES INVOLVING ENZYMES, NUCLEIC ACIDS OR MICROORGANISMS; COMPOSITIONS OR TEST PAPERS THEREFOR; PROCESSES OF PREPARING SUCH COMPOSITIONS; CONDITION-RESPONSIVE CONTROL IN MICROBIOLOGICAL OR ENZYMOLOGICAL PROCESSES
- C12Q2600/00—Oligonucleotides characterized by their use
- C12Q2600/156—Polymorphic or mutational markers
Landscapes
- Chemical & Material Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Organic Chemistry (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Health & Medical Sciences (AREA)
- Engineering & Computer Science (AREA)
- Wood Science & Technology (AREA)
- Zoology (AREA)
- Genetics & Genomics (AREA)
- Molecular Biology (AREA)
- Biotechnology (AREA)
- General Engineering & Computer Science (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Biochemistry (AREA)
- Microbiology (AREA)
- General Health & Medical Sciences (AREA)
- Physics & Mathematics (AREA)
- Biophysics (AREA)
- Immunology (AREA)
- Analytical Chemistry (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Biomedical Technology (AREA)
- Plant Pathology (AREA)
- Measuring Or Testing Involving Enzymes Or Micro-Organisms (AREA)
- General Chemical & Material Sciences (AREA)
Abstract
ESTRATÉGIAS DE SEQUENCIAMENTO DE REGIÃO DE INTERESSE GENÔMICA EM 3D. A invenção refere-se a métodos para determinar a sequência de uma região genômica de interesse que compreende uma sequência de nucleotídeo alvo que compreende fragmentar um DNA reticulado, ligar o DNA reticulado fragmentado, reverter a reticulação e determinar pelo menos parte das sequências de fragmentos de DNA ligados que compreendem uma sequência de nucleotídeo alvo.
Description
A presente invenção refere-se ao campo da biologia molecular e, mais particularmente, à tecnologia de DNA. A invenção se refere em mais detalhes ao sequenciamento de DNA. A invenção se refere a estratégias para determinar (parte de) uma sequência de DNA de uma região genômica de interesse. Em particular, a invenção se refere à determinação da sequência de partes de um genoma que estão em uma configuração espacial entre si. A invenção se refere, adicionalmente, a usos dos métodos da invenção no desenvolvimento de diagnósticos personalizados e tratamento médico, na triagem de tecidos quanto à presença de malignidades e outras afecções.
Um esforço considerável foi dedicado para desenvolver estratégias de "enriquecimento alvo" para sequenciamento, nas quais regiões genômicas de uma amostra de DNA são capturadas seletivamente e/ou amplificadas seletivamente e sequenciadas subsequentemente (revisado em Mamanova et al., Nature Methods, 2010, (2):111 a 118). Estratégias de enriquecimento genômicas são importantes, conforme elas permitem foco em uma região genômica particular, a qual, em comparação a análise de genoma completa, é mais eficaz com custo e tempo, e também muito menos difícil de analisar. Estratégias de enriquecimento genômicas diferentes existem. Por exemplo, realizar uma reação de PCR, com o uso de um único par de iniciador, irá amplificar uma região genômica e, portanto, enriquecer aquela região genômica. Entretanto, o tamanho de produto de PCR que pode ser feito é limitado. Protocolos de PCR longos têm atualmente um limite superior de 10 a 40kB o qual pode ser amplificado (Cheng et al., Proc Natl Acad Sei USA, 1994; 91 (12): 5.695 a 5.699), mas essas abordagens tendem a carecer de robustez e cada PCR requer otimização e validação, e ainda, o limite de tamanho é limitado. A fim de aumentar o tamanho de regiões que podem ser amplificadas, bem como a robustez do ensaio, abordagens com azulejos foram desenvolvidas com o uso de uma multidão de pares de iniciador de PCR projetados especificamente para uma região genômica de interesse. Esses iniciadores são usados, por exemplo, em uma abordagem de PCR de 5 multiplexo ou em um PCR de RainDance. Vários métodos enzimáticos, tal como circularização alvo, são compatíveis com tais estratégias de amplificação direcionadas. Outros métodos envolvem o uso de sondas de captura, em uma armação ou em solução, em que sondas de 60 a 120 bases em comprimento são usadas para capturar a região genômica de interesse por meio de 10 hibridização.
Conforme é claro a partir dos exemplos acima, a fim de enriquecer uma região genômica de interesse, informações de sequência em toda a região genômica de interesse são requeridas antecipadamente, uma vez que isso é necessário para projetar sondas e/ou iniciadores para capturar e/ou 15 amplificar a região genômica de interesse. Por exemplo, enriquecer uma sequência de 30 Mb, 6.000 PCRs separados seriam requeridos tipicamente. Com sondas de captura, ainda mais informações de sequência são requeridas, conforme pelo menos tanto quanto 250.000 sondas de 120bp seriam requeridas e precisam ser projetadas para capturar uma sequência de 30 Mb. 2 o Esses ensaios são parciais ao usar dados de sequência para as sondas e/ou iniciadores os quais cobrem amplamente a região genômica de interesse. Elas não pegam sequência para desviar muito das sequências de modelo projetadas e irão, portanto, por exemplo, não detectar inserções. Adicionalmente, essas abordagens requerem fragmentar DNA em, tipicamente, 2 5 sequências de poucos 100 pares de base antes da análise. Isso significa que a região genômica de interesse é quebrada em muitos pedaços, resultando em perda de informações, entre outros, em relação à redisposições dentro da região de interesse. Por isso, existe uma necessidade para estratégias de enriquecimento genômicas aprimoradas as quais são muito menos parciais, as 30 quais não requerem milhares de sequências curtas, e, as quais habilitam sequenciamento completo neutro de hipótese da região de interesse.
No estudo da arquitetura nuclear de mamífero, os ensaios de capiura de conformação oe cromossomo (3C/4C) foram desenvolvidos, com os quais a organização estrutural de uma região genômica pode ser analisada 5 (WO 2007/004057, WO 2008/08845). Essas tecnologias envolvem a reticulação in vivo de células, por exemplo, com formaldeído, de modo que a arquitetura de cromatina que incluir o DNA seja fixada em sua arquitetura tridimensional. Além disso, a cromatina é fragmentada, por exemplo, com uma enzima de restrição, seguida pela ligação dos fragmentos de DNA reticulados. 10 O resultado é que fragmentos de DNA que estão próximos um do outro são ligados. Os produtos de ligação são subsequentemente amplificados por PCR e analisados para a frequência de interação de fragmentos de DNA ligados, a qual é indicativa da proximidade de fragmentos. A amplificação de PCR pode ser baseada em uma sequência alvo dentro da região genômica de interesse. 15 Uma frequência alta de interação com a região genômica de interesse indica uma proximidade, uma frequência baixa de interação indica uma proximidade distante. A fim de identificar os fragmentos de DNA, as informações de sequência são requeridas. Tais informações de sequência podem ser fornecidas ao detectar fragmentos amplificados com uma microarmação, sendo 20 que compreende sondas, ou por sequenciamento uma parte de fragmentos amplificados pequena (tipicamente, um mínimo de 20 a 30 pares base é suficiente para identificar a posição correspondente em um genoma). De qualquer forma, o número de fragmentos de DNA identificados, isto é, a frequência de interação, indica a proximidade do fragmento ao ponto de vista, 2 5 cujas informações podem ser usadas para determinar interações intracromossômicas ou intercromossômicas.
Foi observado agora que o procedimento de reticulação e fragmentação de DNA dentro de uma célula, e ligação subsequente de 30 fragmentos de DNA reticulados, pode fornecer um ponto de início ideal para analisar uma região genômica de interesse que compreende uma sequência de nucleotídeo alvo, isto é, o modelo de cromossomo linear que cerca a sequência de nucieotíaeo alvo. A invenção e baseada no conceito de que reticuiação de DNA irá reticular preferencialmente aquelas sequências que estão próximas no 5 modelo de cromossomo linear a uma sequência de nucleotídeo alvo.
Formaldeído, por exemplo, pode ser usado como um reticulador. Após reticular, o DNA pode ser submetido a tratamentos (enzimático), isto é fragmentação e ligação, enquanto que o DNA permanece em seu estado reticulado. Somente fragmentos reticulados os quais estão na proximidade um do outro podem ser 10 ligados. Os fragmentos de DNA que se ligam ao fragmento de DNA que compreende a sequência de nucleotídeo alvo são de fato representativos da região genômica de interesse que compreende a sequência de nucleotídeo alvo. Isso ocorre porque a chance de reticuiação intracromossômica está em uma média sempre maior que frequências de reticuiação intercromossômica. 15 Em geral, a chance de fragmentos diferentes serem reticulados correlaciona inversamente com a distância linear. Como uma estimativa, e dependendo das afecções de reticuiação reais, 20 a 30% dos fragmentos ligados com um nucleotídeo alvo de interesse estão localizados dentro de 0,5 Mb da sequência de nucleotídeo alvo, enquanto que 50 a 80% dos fragmentos ligados com um 20 nucleotídeo alvo de interesse originam do cromossomo que compreende a sequência de nucleotídeo alvo. Os fragmentos de DNA ligados que compreendem a sequência de nucleotídeo alvo e, portanto, a região genômica de interesse, podem ser amplificados, isto é, enriquecidos, ao usar um ou mais iniciadores de oligonucleotídeo que reconhece a sequência de nucleotídeo 2 5 alvo. A sequência da região genômica de interesse pode ser determinada subsequentemente com o uso de (rendimento alto) tecnologias de sequenciamento bem conhecidas na técnica. O método é pouco parcial, conforme nenhuma informação extensiva é requerida para focar na região genômica de interesse. Por exemplo, uma região genômica de interesse pode 30 compreender um alelo de interesse. Uma sequência de nucleotídeo alvo pode ser selecionada de modo que não esteja dentro da sequência do alelo de interesse. Uma região genômica de interesse pode então ser amplificada com o uso de uma sequência de nucleotídeo alvo, sem requerer informações de sequência do alelo de interesse. Portanto, o alelo de interesse pode ser enriquecido, sem requerer qualquer sequência daquele alelo. O efeito é que o método de enriquecimento não é parcial ao usar oligonucleotídeos e/ou sondas as quais cobre a sequência alélica de interesse. Adicionalmente, conforme a etapa de ligação envolve os fragmentos que estão próximos um do outro, o método também pode permitir a análise de sequência de alelos separados. Por exemplo, quando uma amostra de DNA reticulada compreende múltiplos alelos (por exemplo, uma vez que a amostra de DNA se origina de uma população de células heterogêneas, ou uma vez que a ploidia é maior que um), cada alelo pode ter uma vizinhança genômica diferente. Um fragmento de DNA, que compreende uma sequência de nucleotídeo alvo, somente irá interagir com fragmentos de DNA que estão no mesmo espaço. Portanto, os fragmentos de DNA ligados são representativos do ambiente genômico a partir do qual os fragmentos originam. Ao determinar pelo menos parte da sequência de todos os fragmentos de DNA ligados diferentes, as sequências de fragmento de DNA podem subsequentemente ser acopladas com o uso das informações de sequência dos fragmentos de DNA ligados diferentes e uma sequência para regiões genômicas separadas de interesse pode ser construída.
Na seguinte descrição e exemplos, diversos termos são usados. A fim de fornecer uma compreensão clara e consistente do relatório descritivo e reivindicações, que incluem o escopo para ser dado tais termos, as seguintes definições são fornecidas. Salvo indicação em contrário no presente documento, todos os termos científicos e técnicos usados têm o mesmo significado conforme compreendido comumente por uma pessoa de habilidade comum na técnica para a esta invenção pertence. As revelações de todas as publicações, pedidos de patente, patentes e outras referências são incorporadas no presente documento em sua totalidade a título de referência.
Métodos para realizar as técnicas convencionais usadas nos métodos da invenção serão evidentes para o trabalhador versado. A prática de técnicas convencionais em biologia molecular, bioquímica, química 5 computacional, cultura de célula, DNA recombinante, bioinformáticas, genômicas, sequenciamento e campos relacionados são bem conhecidos para aqueles versados na técnica e são discutidos, por exemplo, nas seguintes referências de literatura: Sambrook et al. ., Molecular Cloning. A Laboratory Manual, 2a Edição, Cold Spring Harbor Laboratory Press, Cold Spring Harbor, 10 N. Y., 1989; Ausubel et al., Current Protocols in Molecular Biology, John Wiley & Sons, New York, 1987 e atualizações periódicas; e a série Methods in Enzymology, Academic Press, San Diego.
Conforme usado no presente documento, as formas singulares "um," "uma" e "o" incluem referentes plurais a menos que o contexto dite 15 claramente o contrário. Por exemplo, um método para isolar "uma" molécula de DNA, conforme usado acima, inclui isolar uma pluralidade de moléculas (por exemplo, dezenas, centenas, milhares, dezenas de milhares, centenas de milhares, milhões, ou mais moléculas).
Uma "região genômica de interesse" de acordo com a invenção é 2 0 uma sequência de DNA de um organismo do qual é desejável determinar, pelo menos parte da sequência de DNA. Por exemplo, uma região genômica a qual é suspeita de compreender um alelo associado com uma doença pode ser uma região genômica de interesse. Conforme usado no presente documento, o termo "alelo(s)" significa qualquer dentre uma ou mais formas alternativas de 2 5 um gene em um local particular. Em uma célula diploide de um organismo, alelos de um dado gene estão localizados em uma localização específica, ou local (locais, no plural) em um cromossomo. Um alelo está presente em cada cromossomo do par de cromossomos homólogos. Portanto, em uma célula diploide, dois alelos e, portanto, duas regiões genômicas separadas 3 0 (diferentes) de interesse podem existir.
Um "ácido nucleico" de acordo com a presente invenção pode incluir qualquer polímero ou oligômero de bases de purina e pirimidina, preferenciaimente citosma, timina, e uracila, e adenina e guanina, respectivamente (Vide Albert L. Lehninger, Principles of Biochemistry, em 793 a 5 800 (Worth Pub. 1982) o qual está incorporado no presente documento em sua totalidade para todos os propósitos). A presente invenção contempla qualquer deoxiribonucleotídeo, ribonucleotídeo ou componente de ácido nucleico de peptídeo, e quaisquer variantes químicas dos mesmos, tal como formas metiladas, hidroximetiladas ou glicolisadas dessas bases, e similares. Os 10 polímeros ou oligômeros podem ser heterogêneos ou homogêneos na composição, e podem ser isolados de fontes de ocorrência natural ou podem ser produzidas artificial ou sinteticamente. Adicionalmente, os ácidos nucleicos podem ser DNA ou RNA, ou uma mistura dos mesmos, e podem existir permanente ou transicionalmente em uma forma de filamento único ou 15 filamento duplo, que inclui estados homoduplex, heteroduplex, e híbridos.
Um “DNA de amostra” é uma amostra que é obtida a partir de um organismo ou de um tecido de um organismo, ou de tecido e/ou cultura de célula, a qual compreende DNA. Um DNA de amostra de um organismo pode ser obtido a partir de qualquer tipo de organismo, por exemplo, 20 microrganismos, vírus, plantas, fungos, animais, humanos e bactérias, ou combinações dos mesmos. Por exemplo, uma amostra de tecido de um paciente humano suspeito de uma infecção bacteriana e/ou infecção virai pode compreender células humanas, mas também vírus e/ou bactérias. A amostra pode compreender células e/ou núcleo de célula. O DNA de amostra pode ser 2 5 de um paciente ou uma pessoa a qual pode estar em risco ou suspeita de ter uma doença particular, por exemplo, câncer ou qualquer outra afecção a qual requer a investigação do DNA do organismo.
Com "reticuiação" de acordo com a invenção é significado reagir DNA em duas posições diferentes, de modo que essas duas posições 30 diferentes possam ser conectadas. A conexão entre essas duas posições diferentes pode ser diretamente, formando uma ligação covalente entre filamentos de DNA. Dois filamentos de DNA podem ser reticulados diretamente com o uso da irradiação de UV, formando ngaçoes covalentes diretamente entre filamentos de DNA. A conexão entre as duas posições diferentes pode 5 ser indiretamente, por meio de um agente, por exemplo, uma molécula de reticulador. Uma primeira seção de DNA pode ser conectada a um primeiro grupo reativo de uma molécula de reticulador que compreende dois grupos reativos, aquele segundo grupo reativo da molécula de reticulador pode ser conectado a uma segunda seção de DNA, sendo que, através disso, reticula a 10 primeira e segunda seção de DNA indiretamente por meio da molécula de reticulador. Uma reticulação também pode ser formada indiretamente entre dois filamentos de DNA por meio de mais que uma molécula. Por exemplo, uma molécula de reticulador típica que pode ser usada é formaldeído. O formaldeído induz reticulações de proteína-DNA e proteína-proteína. O 15 formaldeído pode, portanto, reticular filamentos de DNA diferentes entre si por meio de suas proteínas associadas. Por exemplo, formaldeído pode reagir com uma proteína e DNA, conectando uma proteína e DNA por meio da molécula de reticulador. Por isso, duas seções de DNA podem ser reticuladas com o uso de formaldeído que forma uma conexão entre uma primeira seção de DNA e 20 uma proteína, a proteína pode formar uma segunda conexão com outra molécula de formaldeído que se conecta a uma segunda seção de DNA, formando, portanto, uma reticulação a qual pode ser retratada como DNA1- reticulador-proteína-reticulador-DNA2. De qualquer forma, é compreendido que a reticulação de acordo com a invenção envolve formar conexões (direta ou 2 5 indiretamente) entre filamentos de DNA que estão em proximidade física um do outro. Os filamentos de DNA podem estar em proximidade física um do outro na célula, conforme DNA é altamente organizado, enquanto que é separado de um ponto de vista de sequência, por exemplo, por 1OOkb. Contanto que o método de reticulação seja compatível com fragmentação subsequente e 30 etapas de ligação, tal reticulação pode ser contemplado para o propósito da invenção.
Uma "amostra de DNA reticulado" é um DNA de amostra o qual foi submetido a feücuiação. A reticulação do DNA de amostra tem o efeito de que o estado tridimensional do DNA dentro da amostra permanece em grande 5 parte intacto. Dessa forma, os filamentos de DNA que estão em proximidade física entre si permanecem próximos um do outro.
"A reticulação reversa", de acordo com a invenção, compreende quebrar as reticulações de modo que o DNA que foi reticulado não seja mais reticulado e é adequado para etapas de amplificação e/ou sequenciamento 10 subsequentes. Por exemplo, realizar um tratamento de K protease em um DNA de amostra que foi reticulado com formaldeído irá digerir a proteína presente na amostra. Uma vez que o DNA reticulado é conectado indiretamente por meio de proteína, o tratamento de protease por si só pode reverter a reticulação entre o DNA. Entretanto, os fragmentos de proteína que permanecem 15 conectados ao DNA pode dificultar amplificação e/ou sequenciamento subsequente. Por isso, reverter as conexões entre o DNA e a proteína pode resultar em “reticulação reversa". A conexão de proteína-reticulado-DNA pode ser revertida através de uma etapa de aquecimento, por exemplo, ao incubar a 70° C. Conforme em um DNA de amostra quantidades grandes de proteína de 2 0 proteína estão presente, é desejável frequentemente diferir a proteína com uma protease em adição. Por isso, qualquer método "reticulação reversa" pode ser contemplado, em que os filamentos de DNA que são conectados em uma amostra reticulada se tornam adequados para sequenciamento e/ou amplificação. 2 5 "Fragmentar DNA" inclui qualquer técnica que, quando aplicada ao DNA, o qual pode ser DNA reticulado ou não, ou qualquer outro DNA, resulta em fragmentos de DNA. Técnicas bem conhecidas na técnica são sonicação, cisalhamento e/ou restrição enzimática, mas outras técnicas também podem ser consideradas. 3 0 Uma "endonuclease de restrição" ou "enzima de restrição" é uma enzima que reconhece uma sequência de nucleotídeo específica (sítio de reconhecimento) em uma molécula de DNA de filamento duplo, e irá clivar ambus us fiiamentos da moiécuia de UNA em ou quase todo sitio de reconhecimento, deixando uma extremidade 3’ ou 5’ brusca ou saliente. A 5 sequência de nucleotídeo específica a qual é reconhecida pode determinar a frequência de clivagem, por exemplo, uma sequência de nucleotídeo de 6 nucleotídeos ocorre em média a cada 4.096 nucleotídeos, em que uma sequência de nucleotídeo de 4 nucleotídeos ocorre com muito mais frequência, em média a cada 256 nucleotídeos. 10 "Ligação", de acordo com a invenção, envolve a união de fragmentos de DNA separados. Os fragmentos de DNA podem ser com extremidades abruptas, ou podem ser cadeias secundárias compatíveis (cadeias secundárias pegajosas) de modo que as saliências possam se hibridizar uma à outra. A união dos fragmentos de DNA pode ser enzimática, 15 com uma enzima de ligase, ligase de DNA. Entretanto, uma ligação não enzimática também pode ser usada, desde que os fragmentos de DNA sejam unidos, isto é, formando uma ligação covalente. Tipicamente uma ligação fosfodiéster entre o grupo fosfato e hidroxila dos filamentos separados é formada. 2 0 Os “iniciadores de oligonucleotídeo”, em geral, se referem a filamentos de nucleotídeos os quais podem iniciar a síntese de DNA. A polimerase de DNA não pode sintetizar DNA de novo sem iniciadores. Um iniciador se hibridiza ao DNA, isto é pares base são formados. Os nucleotídeos que podem formar pares base, que são complementares entre si, são, por 2 5 exemplo, citosina e guanina, timina e adenina, adenina e uracila, guanina e uracila. A complementaridade entre o iniciador e o filamento de DNA existente não precisa ser 100%, isto é, nem todas as bases de um iniciador precisam ser par base com o filamento de DNA existente. Da extremidade 3' de um iniciador hibridizado ao filamento de DNA existente, nucleotídeos são incorporados com 30 o uso do filamento existente como um modelo (síntese de DNA direcionada por modelo). Podemos nos referir às moléculas de oligonucleotídeo sintéticas as quais são usadas em uma reação de amplificação como "iniciadores".
"Ampiificação" se refere a uma reação de amplificação de polinucleotídeo, a saber, uma população de polinucleotídeos que são 5 replicados de uma ou mais sequências de início. A amplificação pode se referir a uma variedade de reações de amplificação, sendo que inclui, mas não é limitada a reação em cadeia de polimerase (PCR), reações de polimerase linear, amplificação baseada em sequência de ácido nucleico, amplificação por círculo rolante e reações similares. 10 "Sequenciamento" se refere a determinar a ordem de nucleotídeos (sequências base) em uma amostra de ácido nucleico, por exemplo, DNA ou RNA. Muitas técnicas estão disponíveis tal como sequenciamento de Sanger e tecnologias de sequenciamento de rendimento alto tal como oferecido por Roche, Illumina e Applied Biosystems. 15 O termo "contíguo" é usado em conexão com a análise de sequência de DNA, e se refere a trechos contíguos remontados de DNA derivados de dois ou mais fragmentos de DNA que têm sequências de nucleotídeo contíguas. Portanto, um contíguo pode ser um conjunto de fragmentos de DNA superpostos que fornecem uma sequência contígua 2 0 (parcial) de uma região genômica de interesse. Um contíguo também pode ser um conjunto de fragmentos de DNA que, quando alinhados a uma sequência de referência, pode formar uma sequência de nucleotídeo contígua. Por exemplo, o termo "contíguo" engloba uma série de (ligados) fragmento(s) de DNA os quais são ordenados em tal forma a ter sobreposição de sequência de 2 5 cada (ligado) fragmento(s) de DNA com pelo menos um de seus vizinhos. O(s) fragmento(s) de DNA ligado(s) ou acoplado(s) (ligado(s)) pode(m) ser ordenado(s) ou manualmente ou, de preferência com o uso de programas de computador apropriados tal como FPC, PHRAP, CAP3 etc., e também pode(m) ser agrupado(s) em contíguos separados. 30 Um "adaptador" é uma molécula de oligonucleotídeo com filamento duplo curta com um número limitado de pares base, por exemplo, cerca de 10 a cerca de 30 pares base em comprimento, os quais são projetados de modo que eles possam ser ligados às extremidades de fragmentos. Os adaptadores são compostos geralmente de dois 5 oligonucleotídeos sintéticos os quais têm sequências de nucleotídeo as quais são parcialmente complementares entre si. Quando misturando os dois oligonucleotídeos sintéticos em solução sob condições apropriadas, eles irão fortalecer um ao outro formando uma estrutura com filamento duplo. Após fortalecimento, uma extremidade da molécula de adaptador será projetada de 10 modo que seja compatível com a extremidade de um fragmento de restrição e pode ser ligada a isso; a outra extremidade do adaptador pode ser projetada de modo que não possa ser ligada, mas isso não precisa ser o caso, por exemplo, quando um adaptador deve ser ligado in entre fragmentos de DNA.
Um "identificador" é uma sequência curta que pode ser 15 adicionada a um adaptador ou um iniciador ou incluído em sua sequência ou usado de outra forma como marcação para fornecer um identificador único. Tal identificador de sequência (ou etiqueta) pode ser uma sequência base única de variação, mas comprimento definido, tipicamente de 4 a 16 bp usado para identificar uma amostra de ácido nucleico específica. Por exemplo, etiquetas de 20 4 bp permitem 4(exp4) = 256 etiquetas diferentes. Os exemplos típicos são sequência de ZIP, conhecidas na técnica como etiquetas usadas comumente para detecção única por hibridização (lannone et al. Cytometry 39:131 a 140, 2000). Os identificadores são úteis de acordo com a invenção, como ao usar tal identificador, a origem de uma amostra (PCR) pode ser determinada mediante 2 5 processamento adicional. No caso de combinação de produtos processados originados de amostras de ácido nucleico diferentes, as amostras de ácido nucleico diferentes podem ser identificadas com o uso de identificadores diferentes. Por exemplo, como de acordo com a invenção, o sequenciamento pode ser realizado com o uso de sequenciamento de rendimento alto, múltiplas 3 0 amostras podem ser combinadas. Os identificadores podem então auxiliar as sequência correspondentes às amostras diferentes. Os identificadores também podem ser incluídos em adaptadores para ligação a fragmentos de DNA que auxiiiam na identificação de sequências de fragmento de DNA. Os identificadores diferem preferencialmente um do outro por pelo menos dois 5 pares base e preferencialmente não contêm duas bases consecutivas idênticas para evitar erros de leitura. A função de identificador pode, algumas vezes, ser combinada com outras funcionalidades tal como adaptadores ou iniciadores.
"Seleção de tamanho" de acordo com a invenção envolve técnicas com as quais faixas de tamanho particular de moléculas, por exemplo, 10 fragmentos de DNA (ligados) ou fragmentos de DNA amplificados (ligados), são selecionados. As técnicas que podem ser usadas para eletroforese de gel de caso, exclusão de tamanho, cromatografia de extração de gel, mas não são limitadas a isso, desde que moléculas com um tamanho particular possam ser selecionadas, tal como técnica será suficiente. 15 Com o termo "alinhando" e "alinhamento" é significado a comparação de duas ou mais sequências de nucleotídeo com base na presença de trechos longos ou curtos de nucleotídeos idênticos ou semelhantes. Os métodos e programas de computador para alinhamento são bem conhecidos na técnica. Um programa de computador o qual pode ser 2 0 usado ou adaptado para alinhar é "Alinhar 2", com autoria de Genentech, Inc., a qual foi depositada com documentação de usuário no Escritório de Direitos Autorais dos Estados Unidos, Washington, D.C. 20559, em 10 de dezembro de 1991.
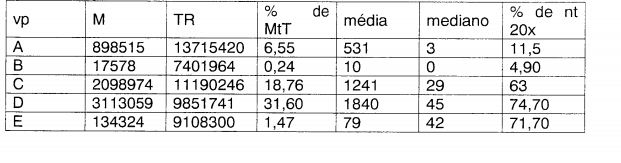
Figuras 2 5 A Figura 1 mostra um esquemático de um método para determinar a sequência de uma região genômica de interesse de acordo com a invenção. O método envolve: (a) reticulação, em que, por exemplo, fixação de formaldeído reticula sequências de DNA próximas espacialmente no núcleo (N) 3 0 (frequentemente sendo sequência próximas no cromossomo (Ch), por exemplo, sequências do mesmo gene) por meio de suas proteínas associadas (por exemplo histonas). 5 fragmentos hipotéticos de uma região genômica de interesse A, b, (J, L) e E são indicados; (b) além disso, o DNA reticulado de amostra é fragmentado, por exemplo, ao realizar uma digestão com uma enzima de restrição (por exemplo um cortador frequente (quatro) (por exemplo, Nlalll)); (c) fragmentos de restrição reticulados são ligados para formar círculos de DNA; (d) após reverter a reticuiação, uma etapa de amplificação, por exemplo, PCR, é realizada com conjunto de iniciador de PCR (inverso) para um ponto de vista próximo a ou dentro da região genômica de interesse. Os fragmentos (A, B, C, D e E) reticulados a esse ponto de vista são reticulados e enriquecidos sobre o remanescente do genoma. Os fragmentos amplificados são sequenciados, por exemplo, sequenciamento através de círculos inteiros (leituras longas), material amplificado por PCR também pode ser primeiro fragmentado para criar uma biblioteca de sequenciamento compatível, por exemplo, sequenciamento de Illumina ou SOLID, (e) além disso, um contíguo é construído a partir das leituras, as sequências podem ser comparadas a um genoma de referência para identificar variação genética. A Figura 2 mostra um esquema do gene BRCA1 gene com 5 pontos de vista diferentes (A, B, C, D e E). A seta preta indica a direção de senso. Os números nos círculos com as setas indicam a posição na sequência de gene. O ponto de vista E está no início do gene e ponto de vista A no fim. Os pontos de vista são separados por aproximadamente 15 a 25 kB. A eletroforese de gel da Figura 3 de amostras de DNA é mostrada tomada durante a separação de um DNA reticulado de amostra para o sequenciamento do gene BRCA1 conforme descrito nos exemplos. (A) coluna M indica um DNA de marcador de Pstil de DNA lambda, a coluna 1 mostra um controle não digerido, a coluna 2 mostra um primeiro controle digerido de Nlalll, a coluna 3 é um controle de ligação após a iigaçao de primeira amostra digerida de Nlalll, a coluna 4 mostra uma segunda digestão com Nspl. 5 (B) a coluna M mostra um marcador de Pstl de DNA lambda. As colunas A, B, C, D e E mostram os produtos de amplificação das amplificações de DNA diferentes, correspondente a amostras da etapa 67 conforme descrito na seção de exemplo, e correspondendo aos pontos de vista descritos na figura 2.
De acordo com um aspecto da invenção, um método é fornecido para determinar a sequência de uma região genômica de interesse que compreende uma sequência de nucleotídeo alvo, que compreende fragmentar um DNA reticulado, ligar o DNA reticulado fragmentado, reverter a reticulação e 15 determinar pelo menos parte da sequência de fragmentos de DNA ligados os quais compreendem uma sequência de nucleotídeo alvo, e usar as sequências determinadas para construir uma sequência da região genômica de interesse.
Uma amostra de DNA reticulado compreende um DNA de amostra o qual foi submetido a reticulação. A reticulação do DNA de amostra 20 conforme está presente nos resultados de amostra que em grande parte mantém a arquitetura tridimensional do DNA. Por exemplo, um agente reticulado padrão que pode ser usado é formaldeído. As amostras podem ser tomadas de um paciente e/ou de tecido de doenças, e também podem ser derivados de outros organismos ou de seções separadas do mesmo 2 5 organismo, tal como amostras de um paciente, uma amostra do tecido saudável e uma amostra do tecido com doença. As amostras podem, portanto, ser analisadas de acordo com a invenção e comparadas com uma amostra de referência, ou amostras diferentes podem ser analisadas e comparadas uma com a outra. Por exemplo, de um paciente que é suspeito de ter câncer de 30 mama, uma biópsia pode ser obtida do tumor suspeito. Outra biopsia pode ser obtida do tecido não doente. A partir de ambas as biópsias de tecido podem ser analisadas de acordo com a invenção. As regiões genômicas de interesses pode ser o gene BHCA1 e BRCA2, cujos genes tem comprimento de 83 e 86 kb (revisado em Mazoyer, 2005, Human Mutation 25:415 a 422). Ao determinar 5 a sequência de região genômica de interesse de acordo com a invenção e comparar as sequências de região genômica das biópsias diferentes entre si e/ou com uma sequência de gene BRCA de referência, mutações genéticas podem ser encontradas que podem auxiliar no diagnóstico do paciente e/ou determinar tratamento do paciente e/ou prever prognóstico de progressão de 10 doença.
Ao fragmentar uma amostra de DNA reticulado, os fragmentos de DNA que se originam de uma região genômica de interesse permanecem próximos um do outro uma vez que são reticulados. Quando esses fragmentos reticulados de DNA são ligados subsequentemente, os fragmentos de DNA da 15 região genômica de interesse, os quais são próximos entre si devido às reticulações, são ligados. Esse tipo de ligação também pode ser referido como ligação de proximidade. Os fragmentos de DNA que compreendem a sequência de nucleotídeo alvo podem ligar com fragmentos de DNA dentro de uma distância linear grande em nível de sequência. Ao determinar (pelo menos 2 0 parte de) a sequência de fragmentos ligados que compreende a sequência de nucleotídeo alvo, as sequências de fragmentos de DNA dentro do entorno espacial da região genômica de interesse são obtidas. Cada sequência de nucleotídeo alvo individual é provável ser reticulado a múltiplos outros fragmentos de DNA. Como uma consequência, frequentemente mais que um 2 5 fragmento de DNA pode se ligado a um fragmento que compreende a sequência de nucleotídeo alvo. Ao combinar sequências (parciais) dos fragmentos (amplificados) de DNA ligados que foram ligados com um fragmento que compreende a sequência de nucleotídeo alvo, uma sequência da região genômica de interesse pode ser construída. Um fragmento de DNA 30 ligado com o fragmento que compreende a sequência de nucleotídeo alvo inclui qualquer fragmento o qual pode estar presente em fragmentos de DNA ligados.
Métodos são conhecidos na técnica que envolvem reticulação de DNA, oem como fragmentação e ligação dos fragmentos de DNA (por exemplo WO 2007/004057 ou WO 2008/08845). Tais métodos são almejados na identificação de frequências de interação entre fragmentos de DNA diferentes, não na identificação da sequência de nucleotídeo primária de fragmentos vizinhos a uma sequência de nucleotídeo alvo. A ideia original de usar 4C para a detecção de frequências de interação somente exigiu uma leitura de sequência curta. A frequência das leituras de sequência curta interagindo são plotadas contra posições cromossômicas das leituras. O padrão de tal trama é indicativo de se uma região genômica particular de interesse pode interagir com uma região em outro lugar no genoma, ou que, por exemplo, translocações entre cromossomos ocorreram. Por exemplo, no caso de uma frequência alta de leituras ser observada em um cromossomo diferente do cromossomo que contém a sequência de nucleotídeo alvo é indicativo de uma translocação. Na invenção atual, a frequência de interação não é determinada. Na invenção atual foi observado que ao fragmentar DNA reticulado e subsequentemente ligar os fragmentos de DNA, de fato uma região genômica que cerca a sequência de nucleotídeo alvo é capturada, que quando sequenciada, permite reconstruir contíguos da região genômica. Em que nos métodos conhecidos na técnica, o foco foi em determinar a frequência de interação de leituras de sequência curta com uma sequência de nucleotídeo alvo, o foco da invenção atual é na determinação da completa, ou pelo menos uma parte grande da sequência de fragmentos de DNA ligados (que compreende o fragmento de DNA com o nucleotídeo alvo) de modo que a partir das sequências de fragmentos de DNA e acoplamentos de fragmentos de DNA ligados, contíguos possam ser construídos para uma região genômica de interesse.
Fragmentos Ligados Linearizados Em uma modalidade da invenção, um método é fornecido para determinar a sequência de uma região genômica de interesse que compreende uma sequência de nucleotídeo alvo, sendo que compreende as etapas de: a) tornecer uma amostra de DNA reticulado: b) fragmentar o DNA reticulado; c) ligar o DNA reticulado fragmentado; d) reverter a reticuiação; e) fragmentar opcionalmente o DNA da etapa d), preferencialmente com uma enzima de restrição; f) ligar opcionalmente o DNA fragmentado da d) ou e) para pelo menos um adaptador; g) amplificar opcionalmente o DNA da etapa d) ou e) que compreende a sequência de nucleotídeo alvo com o uso de pelo menos um iniciador de oligonucleotídeo o qual se hibridiza à sequência de nucleotídeo alvo, ou amplificar o DNA da etapa f) com o uso de pelo menos um iniciador adicional o qual se hibridiza ao pelo menos um adaptador; h) determinar pelo menos parte da sequência dos fragmentos (amplificados) de DNA ligados da etapa d), e), f) ou g) que compreende a sequência de nucleotídeo alvo de preferência com o uso de sequenciamento de rendimento alto; i) construir um contíguo da região genômica de interesse a partir das sequências determinadas. Na etapa a) uma amostra de DNA reticulado é fornecida conforme destacado no presente documento em outro lugar.
A amostra de DNA reticulado é fragmentada na etapa b). Ao fragmentar o DNA reticulado, fragmentos de DNA são produzidos os quais são mantidos juntos pelas reticulações. A etapa de fragmentação b) pode compreender sonicação, e pode ser seguida pelo reparo de extremidade de DNA enzimático. Os resultados de sonicação na fragmentação de DNA em sítios aleatórios, os quais podem ser com extremidades abruptas, ou podem ter cadeias secundárias de 3' ou 5', conforme esses pontos de quebra de DNA ocorrem aleatoriamente, o DNA pode ser reparado (enzimaticamente), preenchendo cadeias secundárias de 31 ou 5', de modo que fragmentos de DNA sejam obtidos os quais têm extremidades abruptas que permitem ligação dos fragmentos a adaptadores e/ou entre si na etapa subsequente c). Alternativamente, as saliências também podem ser feitas com extremidades abruptas ao remover nucleotídeos de cadeias secundárias, com o uso de, por exemplo, exonucleases. A etapa de fragmentação b) também pode compreender fragmentação com uma ou mais enzimas de restrição, ou combinações das mesmas. A fragmentação com uma enzima de restrição é vantajosa, conforme pode permitir controle do tamanho de fragmento médio. Os fragmentos que são formados podem ter cadeias secundárias compatíveis ou extremidades abruptas que permitem ligação dos fragmentos na etapa subsequente c). Ademais, quando se divide uma amostra de DNA reticulado em uma pluralidade de subamostras, para cada subamostra de enzimas de restrição de subamostra com sítios de reconhecimento diferentes podem ser usadas. Isso é vantajoso porque ao usar enzimas de restrição diferentes que têm sítios de reconhecimento diferentes, fragmentos de DNA diferentes podem ser obtidos a partir de cada subamostra.
Na próxima etapa c), os fragmentos são ligados. Visto que um fragmento que compreende uma sequência de nucleotídeo alvo pode ser reticulado para outros fragmentos de DNA, mais que um fragmento de DNA pode ser ligado ao fragmento que compreende a sequência de nucleotídeo alvo. Isso pode resultar em combinações de fragmentos de DNA os quais estão próximos uns dos outros conforme eles são mentidos juntos pelas reticulações. Combinações e/ou ordem diferentes dos fragmentos de DNA em fragmentos de DNA ligados podem ser formadas. No caso dos fragmentos de DNA serem obtidos por meio de restrição enzimática, o sítio de reconhecimento da enzima de restrição é conhecido, o qual torna possível identificar os fragmentos conforme restos de ou sítios de reconhecimento de enzima de restrição reconstituída podem indicar a separação entre fragmentos de DNA diferentes. No caso dos fragmentos de DNA serem obtidos por meio de fragmentação aleatória, tal como sonicação e reparo de extremidade de DNA enzimática subsequente, pode ser mais difícil distinguir um fragmento de outro, mdependentemente de qual método de fragmentação está sendo usado, a etapa de ligação c) pode ser realizada na presença de um adaptador, 5 sequências de adaptador de ligação entre fragmentos. Alternativamente o adaptador pode ser ligado em uma etapa separada. Isso é vantajoso porque os fragmentos diferentes podem ser facilmente identificados ao identificar as sequências de adaptador as quais estão localizadas entre os fragmentos. Por exemplo, no caso de extremidades de fragmento de DNA serem extremidades 10 abruptas, a sequência de adaptador seria adjacente a cada uma das extremidades de fragmento de DNA, indicando o limite entre fragmentos de DNA separados. Além disso, a reticulação é revertida na etapa d), a qual resulta em um agrupamento de fragmentos de DNA ligados que compreendem dois ou mais fragmentos. Uma subpopulação do agrupamento de fragmentos 15 de DNA ligados compreende um fragmento de DNA o qual compreende a sequência de nucleotídeo alvo. Ao reverter a reticulação, a fixação estrutural/espacial do DNA é liberada e a sequência de DNA se torna disponível para etapas subsequentes, por exemplo, amplificação e/ou sequenciamento, conforme DNA reticulado pode não ser adequado para tais 2 0 etapas. As etapas subsequentes e) e/ou f) podem ser realizadas após a reversão da reticulação, entretanto, as etapas e) e/ou f) também podem ser realizadas enquanto que os fragmentos de DNA ligados ainda estão no estado reticulado.
Os fragmentos de DNA ligados podem opcionalmente ser 2 5 fragmentados na etapa e), preferencialmente com uma enzima de restrição. A primeira etapa de fragmentação e a segunda etapa de fragmentação opcional podem ser destinadas a obter fragmentos de DNA ligados de um tamanho o qual é compatível com a etapa de amplificação e/ou etapa de determinação de sequência. Adicionalmente, uma segunda etapa de fragmentação, 30 preferencialmente com uma enzima, pode resultar em extremidades de fragmento ligadas as quais são compatíveis com a ligação opcional de um adaptador na etapa f). A segunda etapa de fragmentação pode ser realizada após reverter a reticuiação, entretanto, também é possível realizar a segunda etapa de fragmentação e) e/ou etapa de ligação f), embora os fragmentos de 5 DNA ainda estejam reticulados.
No caso de a etapa de fragmentação b) e e) compreenderem enzimas de restrição, é preferencial que o sítio de reconhecimento de enzima de restrição da etapa e) seja maior que o sítio de reconhecimento da etapa b). A enzima de e), portanto, corta em uma frequência inferior à etapa b). Isso 10 significa que o tamanho de fragmento de DNA médio da etapa b) é menor que o tamanho de fragmento médio da etapa e) obtido após restringir DNA. Desta forma, na primeira etapa de fragmentação, fragmentos relativamente pequenos são formados, os quais são ligados subsequentemente. Uma vez que a segunda enzima de restrição da etapa e) corta com menos frequência que a 15 etapa b), a maioria dos fragmentos de DNA pode não compreender o sítio de reconhecimento de restrição da etapa e). Portanto, quando os fragmentos de DNA ligados são fragmentados subsequentemente na segunda etapa, muitos dos fragmentos de DNA da etapa b) podem permanecer intactos. Isso é útil porque as sequências combinadas dos fragmentos de DNA da etapa b) podem 2 0 ser usadas para construir um contíguo para a região genômica de interesse. Se a fragmentação da etapa b) for menos frequente que a fragmentação da etapa c), o resultado seria que os fragmentos da etapa b) são fragmentados, os quais podem resultar na perda de sequências de DNA relativamente grandes que são úteis para construir um contíguo. Portanto, independentemente de qual método 2 5 seja usado para fragmentar na etapa b) e e), é preferencial que a fragmentação da etapa b) seja mais frequente em comparação com a etapa e), de modo que os fragmentos de DNA da etapa b) possam permanecer em grande parte intacta, isto é, não em grande parte não fragmentados pela etapa e).
Para os fragmentos de DNA ligados obtidos da etapa d) ou e), 30 pelo menos um adaptador é ligado opcionalmente. As extremidades dos fragmentos de DNA ligados precisam ser compatíveis com ligação de tal adaptador. Uma vez que os fragmentos de DNA ligados da etapa d) ou e) pooem ser DNA linear, a ligação de um adaptador pode fornecer uma sequência de hibridização de iniciador. A sequência de adaptador ligada com 5 fragmentos de DNA ligados que compreendem a sequência de nucleotídeo alvo, irão fornecer moléculas de DNA as quais podem ser amplificados com o uso de PCR.
Na próxima etapa g), o DNA da etapa f) que compreende a sequência de nucleotídeo alvo pode ser amplificado com o uso de pelo menos 10 um iniciador de oligonucleotídeo o qual se hibridiza à sequência de nucleotídeo alvo, e pelo menos um iniciador adicional o qual se hibridiza ao pelo menos um adaptador. Como a etapa f) de ligação de um adaptador é opcional, o DNA da etapa d) ou e) que compreende o nucleotídeo alvo também pode ser amplificado na etapa g) com o uso de pelo menos um iniciador de 15 oligonucleotídeo o qual se hibridiza à sequência de nucleotídeo alvo.
Além disso, a sequência dos fragmentos (amplificados) de DNA ligados obtidos na etapa d), e), f) ou g) que compreende a sequência de nucleotídeo alvo é determinada. A determinação da sequência é preferencialmente realizada com o uso de tecnologia de sequenciamento de 2 0 rendimento alto, como isso é mais conveniente e permite que um número alto de sequências seja determinado para cobrir a região genômica completa de interesse. A partir dessas sequências determinadas um contíguo pode ser construído da região genômica de interesse. Quando sequências dos fragmentos de DNA são determinadas, leituras de cadeias secundárias podem 2 5 ser obtidas a partir da qual a região genômica de interesse pode ser construída. No caso de os fragmentos de DNA serem obtidos por fragmentação aleatória, a natureza aleatória da etapa de fragmentação já pode resultar em fragmentos de DNA os quais quando sequenciados resultam em leituras de cadeias secundárias. Ao aumentar o tamanho de amostra, por exemplo, 30 aumentar o número de células analisadas, a confiabilidade da região genômica de interesse que é construída pode ser aumentada. Alternativamente, quando na etapa b) uma pluralidade de subamostras é analisada, com o uso de enzimas oe restrição diterentes, leituras de cadeia secundária também serão obtidas. Ao aumentar a pluralidade de subamostras, o número de fragmentos 5 de cadeia secundária aumentará, o qual pode aumentar a confiabilidade do contíguo da região genômica de interesse que é construído. A partir dessas sequências determinadas as quais podem superpor, um contíguo pode ser construído. Alternativamente, se as sequências não se sobrepuserem, por exemplo quando uma única enzima de restrição pode ter sido usada na etapa 10 b), o alinhamento de fragmentos (ligados) fragmentos de DNA com uma sequência de referência podem permitir construir um contíguo da região genômica de interesse.
Fragmentos Ligados Circularizados Em uma modalidade alternativa, um método é fornecido para 15 determinar a sequência de uma região genômica de interesse que compreende uma sequência de nucleotídeo alvo, que compreende as etapas de: a) fornecer uma amostra de DNA reticulado; b) fragmentar o DNA reticulado; c) ligar o DNA reticulado fragmentado; d) reverter a reticulação; e) fragmentar opcionalmente o DNA da etapa d), preferencialmente com uma enzima de restrição f) circularizar o DNA da etapa d) ou e); g) opcional ou preferencialmente, amplificar o DNA circularizado 2 5 que compreende a sequência de nucleotídeo alvo que usa preferencialmente pelo menos um iniciador o qual se hibridiza à sequência de nucleotídeo alvo; h) determinar pelo menos parte da sequência dos fragmentos (amplificados) de DNA ligados que compreendem o nucleotídeo alvo que usa sequenciamento de rendimento alto; i) construir um contíguo da região genômica de interesse a partir das sequências determinadas.
Na etapa a), uma amostra de DNA reticulado é fornecida conforme destacado no presente documento em outro lugar. A amostra de DNA reticulado é fragmentada na etapa b). Ao fragmentar o DNA reticulado, os fragmentos de DNA são produzidos os quais são mantidos juntos pelas reticulações. A etapa de fragmentação b) pode compreender sonicação, e podem ser seguidos por reparo de extremidade de DNA enzimático. Os resultados de sonicação na fragmentação de DNA em sítios aleatórios, as quais podem ser extremidades abruptas, ou podem ter cadeia secundárias de 3' ou 5', conforme esses pontos de quebra de DNA ocorrem aleatoriamente, o DNA pode ser reparado (enzimaticamente), preenchendo em cadeias secundárias de 3' ou 5' possíveis, de modo que fragmentos de DNA sejam obtidos os quais têm extremidades abruptas que permitem ligação dos fragmentos para adaptadores ou entre si na etapa subsequente c). Alternativamente, as cadeias secundárias também podem ser feitas extremidades abruptas ao remover nucleotídeos de cadeia secundária, com o uso de, por exemplo, exonucleases. A etapa de fragmentação b) também pode compreender fragmentação com uma enzima de restrição, ou combinações da mesma. A fragmentação com uma enzima de restrição é vantajosa conforme permite controle do tamanho de fragmento médio. Ademais, os fragmentos que são formados terão cadeias secundárias compatíveis ou extremidades abruptas que permitem ligação dos fragmentos na etapa subsequente c) sem exigir modificação adicional. Além disso, quando se divide uma amostra de DNA reticulado em uma pluralidade de subamostras, para cada subamostra enzimas de restrição com sítios de reconhecimento diferentes podem ser usadas. Isso pode ser vantajoso porque ao usar enzimas de restrição diferentes que têm sítios de reconhecimento diferentes, fragmentos de DNA diferentes podem ser obtidos de cada subamostra.
Na próxima etapa c), os fragmentos são ligados. No caso dos fragmentos de DNA serem obtidos por meio de restrição enzimática, o sítio de reconhecimento da enzima de restrição é conhecido, o qual torna possível identificar os fragmentos como restos de ou sítios de reconhecimento de enzima de restrição reconstituída podem indicar a separação entre fragmentos de DNA diferentes. No caso dos fragmentos de DNA serem obtidos por meio de fragmentação aleatória, tal como sonicação e reparo de extremidade de DNA enzimático subsequente, pode ser mais difícil distinguir um fragmento de outro. Independentemente de qual método de fragmentação seja usado, a etapa de ligação c) pode ser realizada na presença de um adaptador, sequências de adaptador de ligação entre fragmentos. Alternativamente o adaptador pode ser ligado em uma etapa separada. Isso é vantajoso porque os fragmentos diferentes podem ser facilmente identificados ao identificar as sequências de adaptador as quais estão entre os fragmentos. Por exemplo, no caso de as extremidades de fragmento de DNA serem extremidades abruptas, a sequência de adaptador seria adjacente às extremidades fragmento de DNA, indicando os fragmentos de DNA separados.
A seguir, a reticulação é revertida na etapa d), a qual resulta em um agrupamento de fragmentos de DNA ligados que compreendem dois ou mais fragmentos. Uma subpopulação do agrupamento de fragmentos de DNA ligados compreende um fragmento de DNA o qual compreende a sequência de nucleotídeo alvo. Ao reverter a reticulação, a fixação espacial /estrutural do DNA é liberada e a sequência de DNA se torna disponível para etapas subsequentes, por exemplo, amplificação e/ou sequenciamento, conforme DNA reticulado pode não ser um substrato adequado para tais etapas. As etapas subsequentes e) e/ou f) podem ser realizadas após a reversão da reticulação, entretanto, as etapas e) e/ou f) também podem ser realizadas enquanto que os fragmentos de DNA ligados ainda estão no estado reticulado.
Os fragmentos de DNA ligados podem opcionalmente ser fragmentados na etapa e), preferencialmente com uma enzima de restrição. A fragmentação pode ser realizada após reverter a reticulação, mas também é considerado que a segunda fermentação é realizada antes da reticulação ser revertida. É preferencial usar uma enzima de restrição para a fragmentação, como uma enzima de restrição permite controle da etapa de fragmentação e resulta, se uma enzima de restrição apropriada for escolhida, em extremidades compatíveis dos fragmentos de DNA ligados que são favoráveis para ligação das extremidades compatíveis, resultando em fragmentos de DNA ligados circularizados, conforme obtido na etapa f). Entretanto, a fragmentação com o uso de outros métodos, por exemplo, cisalhamento e/ou sonicação e reparo de extremidade de DNA enzimático subsequente, de modo que DNA de filamento duplo de extremidades abruptas seja formado também possa ser ligado para formar DNA circularizados.
A primeira etapa de fragmentação e a segunda etapa de fragmentação opcional são destinadas à obtenção de fragmentos de DNA ligados os quais são compatíveis com a circularização subsequente, etapa de amplificação e/ou etapa de determinação de sequência. No caso de a etapa de fragmentação b) e e) compreender enzimas de restrição, é preferencial que a etapa de fragmentação e) resulta em, em média, fragmentos maiores conforme seria obtido na etapa de fragmentação b). No caso de a etapa de fragmentação b) e e) compreender enzimas de restrição, é preferencial que o sítio de reconhecimento de enzima de restrição da etapa e) seja maior que o sítio de reconhecimento da etapa b). A enzima de e), portanto, corta em uma frequência inferior à etapa b). Isso significa que o tamanho de fragmento de DNA médio da etapa b) é menor que o tamanho de fragmento médio da etapa e) obtido após restringir DNA. Isso forma, na primeira etapa de fragmentação, fragmentos relativamente pequenos são formados, os quais são ligados subsequentemente. Como a segunda enzima de restrição da etapa e) corta com menos frequência que a etapa b), a maioria dos fragmentos de DNA pode não compreender o sítio de reconhecimento de restrição da etapa e). Portanto, quando os fragmentos de DNA ligados fragmentados subsequentemente na segunda etapa, muitos dos fragmentos de DNA da etapa b) podem permanecer intactos. Isso é útil uma vez que as sequências combinadas dos fragmentos de
DNA da etapa b) podem ser usadas para construir um contíguo para a região genômica de interesse. Se a fragmentação da etapa b) for menos frequente que a fragmentaçao da etapa c), o resultado seria que os fragmentos da etapa b) são fragmentados, os quais podem resultar na perda de sequências de DNA relativamente grandes que são úteis para construir um contíguo. Portanto, independentemente de qual método seja usado para fragmentar na etapa b) e e), é preferencial que a fragmentação da etapa b) seja mais frequente em comparação com a etapa e), de modo que fragmentos de DNA da etapa b) permaneçam, em grande parte, intactos, isto é, sejam não fragmentados em grande parte pela etapa e).
Os fragmentos de DNA ligados obtidos da etapa d) ou e), dos quais a reticuiação foi revertida, são, a seguir, circularizados na etapa f). Pode ser vantajoso reverter a reticuiação antes da circularização, porque pode ser desfavorável circularizar DNA reticulado enquanto está reticulado. Entretanto, a circularização também pode ser realizada enquanto os fragmentos de DNA ligados estão reticulados. Pode ser possível, ainda, que uma etapa de circularização adicional não seja exigida, já que durante a etapa de ligação, fragmentos de DNA ligados circularizados são prontamente formados, e, portanto, a etapa de circularização f) ocorreria simultaneamente à etapa c). Entretanto, é preferível realizar uma etapa de circularização adicional. A circularização envolve a ligação das extremidades dos fragmentos de DNA ligados de modo que um círculo fechado seja formado. O DNA circularizado que compreende fragmentos de DNA ligados que compreendem a sequência de nucleotídeo alvo pode ser, subsequentemente ampliado como uso de pelo menos um iniciador que hibridiza à sequência de nucleotídeo alvo. Para a etapa de ampliação, reverter a reticuiação é exigido, já que DNA reticulado pode atrasar ou evitar a ampliação. De preferência, dois iniciadores são usados os quais hibridizam na sequência de nucleotídeo alvo em uma reação PCR inversa. Dessa forma, fragmentos de DNA do DNA circularizado, que são ligados ao fragmento de DNA que compreende a sequência de nucleotídeo alvo, podem ser ampliados.
A seguir, a sequência dos fragmentos de DNA ligados (ampliados) obtidos na etapa d), e), t) ou g) que compreende a sequência de nucleotídeo alvo é determinada. A determinação da sequência é, de preferência, realizada 5 com o uso de tecnologia de sequenciamento de alto rendimento, já que isso é mais conveniente e permite que um alto número de sequências seja determinado para cobrir a região genômica completa de interesse. A partir dessas sequências determinadas, um contíguo pode ser construído na região genômica de interesse. Quando as sequências dos fragmentos de DNA são 10 determinadas, a leituras sobrepostas podem ser obtidas a partir das quais a região genômica de interesse pode ser construída. Caso os fragmentos de DNA fossem obtidos por fragmentação aleatória, a natureza aleatória da etapa de fragmentação já pode resultar em fragmentos de DNA que, quando sequenciados, resultam em leituras sobrepostas. Ao aumentar o tamanho de 15 amostra, por exemplo, aumentar o número de células analisadas, a confiabilidade da região genômica de interesse que é construída pode ser aumentada. Alternativamente, quando na etapa b) uma pluralidade de subamostras é analisada, como uso de enzimas de restrição diferentes, leituras sobrepostas também serão obtidas. Ao aumentar a pluralidade de 2 0 subamostras, o número de fragmentos sobrepostos aumentará, o que pode aumentar a confiabilidade do contíguo da região genômica de interesse que é construída. A partir dessas sequências determinadas que podem se sobrepor, um contíguo pode ser construído. Alternativamente, se as sequências não se sobrepuserem, por exemplo, quando uma única enzima de restrição pode ter 2 5 sido usada na etapa b), o alinhamento de fragmentos de DNA (ligados) com uma sequência de referência pode permitir que se construa um contíguo da região genômica de interesse.
Múltiplas sequências alvo Em uma modalidade, um método para determinar a sequência de 30 uma região genômica de interesse que compreende duas sequências de nucleotídeo alvo é fornecido. Esse método pode envolver as mesmas etapas que aquelas determinadas acima até a etapa da ampliação. A etapa de ampliação usa, agora, não uma sequência de nucleotídeo alvo, mas duas. Para as duas sequências de nucleotídeo alvo, dois iniciadores diferentes são usados 5 em uma reação PCR, um iniciador para cada sequência de nucleotídeo alvo. Quando os dois sítios de ligação de iniciador das duas sequências de nucleotídeo alvo estão presentes em um fragmento de DNA ligado, os dois iniciadores ampliarão a sequência entre os sítios de ligação de iniciador contanto que os sítios de ligação de iniciador tenham a orientação correta. Ter 10 um fragmento de DNA ligado circularizado pode ser vantajoso já que a chance de os dois sítios de ligação de iniciador terem a orientação correta é mais alta, se comparada a um fragmento de DNA ligado linear (duas de quatro orientações ampliarão, se comparadas a uma de quatro para um fragmento de DNA ligado linear). Em uma modalidade adicional, além das duas sequências 15 de nucleotídeo alvo, a região genômica de interesse compreende nucleotídeos alvo adicionais, para cada nucleotídeo alvo, um iniciador é usado na reação de ampliação PCR. Combinar múltiplos nucleotídeos alvo e corresponder iniciadores em uma única ampliação aumentará a chance de combinações dos iniciadores produzirem um amplicão. 20 Por exemplo, conforme descrito na seção de exemplo 5, nucleotídeos alvo diferentes foram usados para o gene BRCA1 (consulte, por exemplo, a figura 2). Uma PCR pode ser realizada ao selecionar um iniciador de uma sequência de nucleotídeo alvo (também chamado de ponto de vista), por exemplo, A com outro, B. Ademais, uma PCR pode ser realizada com o uso 2 5 do iniciador de cada sequência de nucleotídeo alvo, A, B, C, D e E. Já que esses nucleotídeos alvo estão em proximidade física em relação uns aos outros, realizar tal ampliação enriquecerá a região genômica de interesse, contanto que os sítios de ligação de iniciador terminem em fragmentos de DNA ligados de modo que um amplicão possa ser gerado. 30 Portanto, os métodos são fornecidos para determinar a sequência de uma região genômica de interesse, de acordo com a invenção, em que a região genômica de interesse compreende, em adição, uma ou mais sequências de nucleotideo alvo e em que, na etapa de ampliação, um iniciador é fornecido o qual hibridiza com a sequência de nucleotídeo alvo e um ou mais iniciadores são fornecidos para um ou mais correspondentes nucleotídeos alvo adicionais, em que os fragmentos de DNA ligados são ampliados, ou DNA circularizado é ampliado, com o uso dos iniciadores.
Determinação da sequência de fragmentos de DNA ligados A etapa de determinação da sequência de fragmentos de DNA ligados compreende, de preferência, sequenciamento de alto rendimento. Métodos de sequenciamento de alto rendimento são bem conhecidos na técnica e, em princípio, qualquer método pode ser contemplado para ser usado na invenção. Tecnologias de sequenciamento de alto rendimento podem ser executadas de acordo com as instruções do fabricante (como, por exemplo, fornecidas por Roche, Illumina or Applied Biosystems). Em geral, adaptadores de sequenciamento podem ser ligados aos fragmentos de DNA ligados (ampliados). Caso o fragmento linear ou circularizado seja ampliado, através do uso, por exemplo, de PCR, conforme descrito no presente documento, o produto ampliado é linear, permitindo a ligação dos adaptadores. Extremidades adequadas podem ser fornecidas para ligar as sequências de adaptador (por exemplo, extremidades abruptas, escalonadas, complementares). Alternativamente, o(s) iniciador(es) usado(s) para PCR ou outro método de ampliação, podem incluir sequências de adaptador, de modo que os produtos ampliados com as sequências de adaptador sejam formados na etapa de ampliação g). Caso o fragmento circularizado não seja ampliado, o fragmento circularizado pode ser fragmentado, de preferência, através do uso, por exemplo, de uma enzima de restrição entre sítios de ligação de iniciador para a reação PCR inversa, de modo que os fragmentos de DNA ligados aos fragmentos de DNA que compreendem a sequência de nucleotídeo alvo permaneçam intactos. Adaptadores de sequenciamento também podem ser incluídos nas etapas c) e f) dos métodos da invenção. Esses adaptadores de sequenciamento podem ser incluídos como parte das sequências de adaptador dos adaptadores ja podem ser opcionalmente usados nessas etapas e/ou adaptadores de sequência separados podem ser fornecidos nessas etapas em adição.
De preferência, leituras longas podem ser geradas no método de sequenciamento de alto rendimento usado. As leituras longas podem permitir a leitura através de múltiplos fragmentos de DNA de fragmentos de DNA ligados. Dessa forma, os fragmentos de DNA da etapa b) podem ser identificados. Sequências de fragmento de DNA podem ser comparados a uma sequência de referência e/ou comparados uns aos outros. Por exemplo, conforme também é explicado daqui em diante, tais sequências de fragmento de DNA podem ser usadas para determinar razão dos fragmentos de células que portam uma mutação genética. Por sequenciamento das sequências de fragmento de DNA de fragmentos de DNA adjacentes a tais sequências, fragmentos de DNA ligados únicos também podem ser identificados. Esse é, em particular, o caso em que fragmentos de DNA foram obtidos na etapa b) por fragmentação aleatória. A chance de duas células fornecerem o exato mesmo fragmento de DNA é muito pequena, quanto mais a de as extremidades de fragmento de DNA às quais tal fragmento está ligado serem iguais. Portanto, ao identificar fragmentos de DNA dessa forma, a razão de células e/ou regiões genômicas de interesse que compreendem uma mutação particular pode ser determinada.
Portanto, não é necessário fornecer uma sequência completa dos fragmentos de DNA ligados. É preferível pelo menos sequenciar através de fragmentos de DNA (múltiplos), de modo que as sequências de fragmento de DNA sejam determinadas.
Também pode ser contemplado ler sequências ainda mais curtas, por exemplo, leituras curtas de 50 a 100 nucleotídeos. Em tal cenário, é preferível fragmentar o DNA ligado (ampliado) em fragmentos menores, que podem ser subsequentemente ligados com um adaptador apropriado adequado para o método de sequenciamento de alto rendimento. Caso um protocolo de sequenciamento padrão seja usado, isso pode significar que as informações em reiação aos fragmentos de DNA ligados podem ser perdidas. Com leituras curtas pode não ser possível identificar uma sequência de fragmento de DNA completa. Caso tais leituras curtas sejam contempladas, pode-se vislumbrar fornecer etapas de processamento adicionais, de modo que fragmentos de DNA ligados separados quando fragmentados sejam ligados ou equipados com identificadores, de modo que, a partir das leituras curtas, os contíguos podem ser construídos para os fragmentos de DNA ligados. Tais tecnologias de sequenciamento de alto rendimento que envolvem leituras de sequência curtas podem envolver sequenciamento de extremidade pareada. Ao usar o sequenciamento de extremidade pareada e leituras de sequência curtas, as leituras curtas de ambas as extremidades de uma molécula de DNA usadas para o sequenciamento, sendo que a molécula de DNA pode compreender fragmentos de DNA diferentes, podem permitir o acoplamento de fragmentos de DNA que foram ligados. Isso ocorre porque duas leituras de sequência podem ser acopladas com o planejamento de uma sequência de DNA relativamente grande em relação à sequência que foi determinada a partir de ambas as extremidades. Dessa forma, os contíguos podem ser construídos para os fragmentos de DNA ligados (ampliados).
Entretanto, o uso de leituras curtas pode ser contemplado sem identificar os fragmentos de DNA, porque a partir das leituras de sequência curtas, uma região genômica de interesse pode ser construída, especialmente quando a região genômica de interesse foi ampliada. Informações em relação aos fragmentos de DNA e/ou região genômica de interesse separada (por exemplo, de uma célula diploide) podem ser perdidas, mas mutações de DNA podem ainda ser identificadas.
Portanto, a etapa para a determinação de pelo menos parte da sequência da sequência de DNA ligado (ampliado) pode compreender leituras de sequência curtas, mas, de preferência, leituras de sequência mais longas são determinadas de modo que as sequências de fragmento de DNA possam ser identificadas. Ademais, também se pode contemplar o uso de estratégias de sequenciamento de alto rendimento diferentes para os fragmentos de DNA ligados (ampliados), por exemplo, combinar leituras de sequência curtas a partir do sequenciamento de extremidade pareada com as extremidades relativamente afastadas com leituras de sequência mais longas, dessa forma, os contíguos podem ser construídos para os fragmentos de DNA ligados (ampliados).
Em uma modalidade, a invenção pode ser usada para fornecer o controle de qualidade de informações de sequência geradas. Na análise das sequências, conforme fornecida por um método de sequenciamento de alto rendimento, erros de sequenciamento podem ocorrer. Um erro de sequenciamento pode ocorrer, por exemplo, durante o alongamento do filamento de DNA, em que a base errada (isto é, não complementar ao modelo) é incorporada no filamento de DNA. Um erro de sequenciamento é diferente de uma mutação, já que o DNA original que é ampliado e/ou sequenciado não compreenderia aquela mutação. De acordo com a invenção, as sequências de fragmento de DNA podem ser determinadas com (pelo menos parcialmente) sequências de fragmentos de DNA ligadas às mesmas, sendo que as sequências podem ser únicas. A característica única dos fragmentos de DNA ligados conforme são formados na etapa c) pode fornecer o controle de qualidade da sequência determinada na etapa h). Quando os fragmentos de DNA ligados são ampliados e sequenciados a uma profundidade o suficiente, cópias múltiplas do(s) mesmo(s) fragmento(s) de DNA único(s) ligado(s) serão sequenciadas. As sequências de cópias que originam do mesmo fragmento de DNA ligado original podem ser comparadas e a ampliação erros de ampliação e/ou sequenciamento podem ser identificados.
Modalidades adicionais Ademais, de acordo com os métodos da invenção, a partir de uma amostra de DNA reticulado, as sequências de múltiplas regiões genômicas de interesse são determinadas. Para cada região genômica de interesse, uma sequência de nucleotídeo alvo é fornecida, para a qual miciador(es)correspondentes podem ser designados. As múltiplas regiões genômicas de interesse podem ser regiões genômicas de interesse que também podem se sobrepor, aumentando, por meio disso, o tamanho da sequência que pode ser determinado. Por exemplo, caso uma sequência de uma região genômica de interesse que compreende uma sequência de nucleotídeo alvo compreenda, tipicamente, 1 MB, combinar regiões genômicas de interesse parcialmente sobrepostas, por exemplo, com uma sobreposição de 0,1 MB, cada uma tendo uma sequência de nucleotídeo alvo correspondente, combinando 5 regiões genômicas de interesse resultaria em uma sequência de 4,6 MB (0,9 + 3* (0,1 + 0,8) + 0,1 + 0,9 = 4,6 MB), estendendo enormemente, por meio disso, o tamanho da região genômica de interesse da qual a sequência pode ser determinada ou, de outra forma, analisada. Múltiplas sequências de nucleotídeo alvo a distâncias definidas em uma região genômica de interesse também podem ser usadas para aumentar a cobertura média e/ou a uniformidade da cobertura através da região genômica.
Ademais, um identificador pode ser incluído em pelo menos um dos iniciadores de oligonucleotídeo da etapa g). Os identificadores também podem ser incluídos nas sequências de adaptador, como as que são usadas para a ligação entre fragmentos durante a etapa de ligação c). Ao incluir um identificador no iniciador de oligonucleotídeo, ao analisar uma pluralidade de amostras ou uma pluralidade de subamostras de DNA reticulado simultaneamente, a origem de cada amostra pode ser facilmente determinada. (Sub)amostras de DNA reticulado podem ter sido processadas diferentemente enquanto a amostra original de DNA reticulado é igual, e/ou amostras de DNA podem ter sido obtidas, por exemplo, de organismos ou pacientes diferentes. Identificadores permitem combinar amostras processadas diferentemente quando o processamento das amostras pode convergir, por exemplo, etapas de procedimento idênticas são realizadas. Tal convergência de processamento pode, em particular, ser vantajosa quando a etapa de sequenciamento h) envolve o sequenciamento de alto rendimento.
Anteriormente a ou após a etapa de ampliação g), de acordo com os métodos da invenção, uma etapa de seleção de tamanho pode ser realizada. Tal etapa de seleção de tamanho pode ser realizada como o uso de cromatografia de extração em gel, eletroforese em gel ou centrifugação de gradiente de densidade, que são métodos, em geral, conhecidos na técnica. De preferência, o DNA é selecionado de um tamanho entre 20 e 20.0000 pares de base, de preferência 50 a 10.0000 pares de base, com máxima preferência, entre 100 e 3.000 pares de base. Uma etapa de separação por tamanho permite a seleção de fragmentos de DNA ligados (ampliados) em uma faixa de tamanho que pode ser ótima para a ampliação por PCR e/ou ótima para o sequenciamento de leituras longas por sequenciamento da próxima geração. O sequenciamento de leituras de 500 nucleotídeos está atualmente comercialmente disponível, avanços recentes por empresas, como a tecnologia Single Molecule Real Time (SMRT™) DNA Sequencing desenvolvida pela Pacific Biosciences (http://www.pacificbiosciences.com/), indicam que leituras de 1,000 a 10.000 nucleotídeos são possíveis.
Caso a ploidia em uma célula de uma região genômica de interesse seja maior que 1, para cada ploidia, um contíguo é construído na etapa h) dos métodos, de acordo com a invenção. Já que o ambiente genômico de qualquer dado sítio alvo no genoma consiste, principalmente, em sequências de genoma de DNA que estão fisicamente perto da sequência alvo no modelo de cromossoma linear, o mesmo permite a reconstrução de cada modelo de cromossoma particular. Caso a ploidia de uma região genômica de interesse seja maior que 1, múltiplas regiões genômicas de interesse estão presentes em uma célula (ou equivalente às mesmas). Essas múltiplas regiões genômicas de interesse não ocupam, em geral, o mesmo espaço, isto é, são separadas em espaço. Quando uma amostra de DNA reticulado de tal célula é fragmentada, a partir de cada região genômica de interesse em uma célula, um fragmento de DNA correspondente que compreende a sequência de nucleotídeo alvo será formado. Esses fragmentos de DNA se ligarão, cada um, a fragmentos de DNA em suas proximidades. Fragmentos de DNA ligados serão, portanto, representativos das diferentes regiões genômicas de interesse. Por exemplo, caso a ploidia seja dois, quando dois fragmentos, cada, que têm uma mutação única e que são separados por 1 MB seriam encontrado juntos em fragmentos de DNA ligados, pode-se concluir que esses dois fragmentos são da mesma região genômica de interesse. Portanto, nesse caso, dois fragmentos foram identificados e ambos são designados à mesma região genômica. Portanto, ao construir um contíguo a partir das sequências dos fragmentos identificados, esses dois fragmentos que portam uma mutação seriam usados para construir um contíguo para uma região genômica particular, enquanto o contíguo construído para a outra região genômica não portaria as mutações.
Deste modo, de acordo com os métodos da invenção, a etapa h) de construção de um contíguo compreende as etapas de: 1) identificar os fragmentos da etapa b); 2) designar os fragmentos a uma região genômica; 3) construir um contíguo para a região genômica a partir das sequências dos fragmentos.
Isso também ocorre quando três fragmentos que compreendem uma única mutação ocorrem (A*, B* e C*) e a ploidia do interesse genômico é dois. Dessa vez, produtos de ligação que compreendem dois dos fragmentos mutacionados são identificados, um produto de ligação que compreende A*B* e um com A*C*. Além disso, produtos de ligação que compreendem fragmentos não mutacionados são identificados como BC e AC. Nesse caso, os fragmentos de DNA ligados A*B e A*C* são acoplados pelo fragmento A* e os fragmentos de DNA ligados BC e AC são acoplados pelo fragmento C. Nesse caso, os fragmentos de DNA A*, B* e C* são designados à mesma região genômica, enquanto A, B e C são designados à outra região genômica.
Portanto, em conformidade, a etapa 2) de designação dos fragmentos a uma região genômica compreende a identificação de produtos de ligação diferentes e o acoplamento dos produtos de ligação diferentes que compreendem os fragmentos de DNA.
Da mesma forma, o mesmo se aplicaria a populações de célula heterogêneas. Por exemplo, caso uma amostra de DNA reticulado seja fornecida a qual compreende uma população de célula heterogênea (por exemplo, células com origens diferentes ou células de um organismo que compreende células normais e células geneticamente mutacionadas (por exemplo, células de câncer)), para cada região genômica de interesse que corresponde a um ambiente genômico diferente (que pode, por exemplo, ser ambientes genômicos diferentes em uma célula ou ambientes genômicos diferentes de células diferentes) contíguos podem ser construídos. Identificação de mutações Em modalidades alternativas, os métodos são fornecidos para a identificação da presença ou ausência de uma mutação genética.
Em uma primeira modalidade, um método é fornecido para identificar a presença ou ausência de uma mutação genética que compreende as etapas a) a h) de qualquer um dos métodos da invenção, conforme descrito acima, em que os contíguos são construídos para uma pluralidade de amostras, que compreendem as etapas de: i) alinhar os contíguos de uma pluralidade de amostras; j) identificar a presença ou ausência de uma mutação genética nas regiões genômicas de interesse a partir da pluralidade de amostras. Alternativamente, um método para identificar a presença ou ausência de uma mutação genética é fornecido, sendo que o método compreende as etapas a) a g) de qualquer um dos métodos da invenção, conforme descrito acima, compreendendo as etapas adicionais de: i) alinhar o contíguo a uma sequência de referência. j) identificar a presença ou ausência de uma mutação genética na região genômica de interesse.
Mutações genéticas podem ser identificadas, por exemplo, ao comparar os contíguos de múltiplas amostras, caso uma (ou mais) das amostras compreendam uma mutação genética, isso pode ser observado, já que a sequência do contíguo é diferente quando comparada à sequência das outras amostras, isto é, a presença de uma mutação genética é identificada. Caso nenhuma diferença de sequência entre contíguos das amostras seja observada, a ausência da mutação genética é identificada. Alternativamente, uma sequência de referência também pode ser usada, à qual a sequência de um contíguo pode ser alinhada. Quando a sequência do contíguo da amostra é diferente da sequência da sequência de referência, uma mutação genética é observada, isto é, a presença de uma mutação genética é identificada. Caso nenhuma diferença de sequência entre o contíguo da amostra ou amostras e a sequência de referência seja observada, a ausência de mutação genética é identificada.
Não é necessário construir um contíguo para identificar a presença ou ausência de uma mutação genética. Contanto que fragmentos de sequências de DNA possam ser alinhados, uns aos outros ou a uma sequência de referência, a presença de ausência de uma mutação genética pode ser identificada. Portanto, em modalidades alternativas da invenção, um método é fornecido para identificar a presença ou ausência de uma mutação genética, de acordo com qualquer um dos métodos, conforme descrito acima, sem a etapa h) de construir um contíguo.
Tal método compreende as etapas a) a g) de qualquer um dos métodos, conforme descrito acima, e as etapas adicionais de: h) alinhar as sequências determinadas dos fragmentos de DNA ligados (ampliados) a uma sequência de referência. i) identificar a presença ou ausência de uma mutação genética nas sequências determinadas. Alternativamente, um método é fornecido para identificar a presença ou ausência de uma mutação genética, em que de uma pluralidade de amostras, as sequências de fragmentos de DNA ligados (ampliados) são determinas, compreendendo as etapas a) a g) de qualquer um dos métodos, conforme descrito acima, compreendendo as etapas adicionais de: h) alinhar as sequências determinadas dos fragmentos de DNA ligados (ampliados) de uma pluralidade de amostras. i) identificar a presença ou ausência de uma mutação genética nas sequências determinadas..
Razão de alelos ou células que portam uma mutação genética Conforme mencionado acima, quando uma amostra de DNA reticulado é fornecida a partir de populações de célula heterogêneas (por exemplo, células com origem diferente ou células de um organismo que compreende células normais e células geneticamente mutacionadas (por exemplo, células de câncer)), para cada região genômica de interesse correspondente a ambientes genômicos diferentes (que podem, por exemplo, ser ambientes genômicos diferentes de alelos diferentes em uma célula ou ambientes genômicos diferentes de células diferentes) contíguos podem ser construídos. Ademais, a razão de fragmentos ou fragmentos de DNA ligados que portam uma mutação genética pode ser determinada, o que pode ser correlacionado à razão de alelos ou células que portam a mutação genética. A ligação de fragmentos de DNA é um processo aleatório, a coleta e ordem de fragmentos de DNA que são parte dos fragmentos de DNA ligados pode ser única e representa uma célula única e/ou uma região genômica de interesse única de uma célula. Ademais, caso a etapa de fragmentação b) compreenda um processo de fragmentação aleatória, como, por exemplo, sonicação, os pontos nos quais o DNA foi rompido podem fornecer uma característica única adicional, especialmente no contexto dos outros fragmentos de DNA aos quais estão ligados (que também podem ter extremidades de fragmento únicas).
Portanto, a identificação de fragmentos de DNA ligados que compreendem o fragmento com a mutação genética também pode compreender a identificação de fragmentos de DNA ligados com uma ordem única a coleta dos fragmentos de DNA. A razão dos alelos ou células que ponam uma mutaçao genetica pode ser importante na avaliação de terapias, por exemplo, em caso de pacientes que estão passando por terapia para câncer. Células de câncer podem portar uma mutação genética particular. A porcentagem de células que portam tal mutação pode ser uma medida para o sucesso ou falha de uma terapia. Em modalidades alternativas, métodos são fornecidos para determinar a razão de fragmentos que portam uma mutação genética, e/ou a razão de fragmentos de DNA ligados que portam uma mutação genética. Nessa modalidade, uma mutação genética é definida como uma mutação genética particular ou uma seleção de mutações genéticas particulares.
Em uma primeira modalidade, um método é fornecido para determinar a razão de fragmentos que portam uma mutação genética a partir de uma população de célula da qual se suspeita ser heteróloga que compreende as etapas a) a h) de qualquer um dos métodos, conforme descrito acima, que compreende as etapas adicionais de: i) identificar os fragmentos da etapa b); j) identificar a presença ou ausência de uma mutação genética nos fragmentos; k) determinar o número de fragmentos que portam a mutação genética; l) determinar o número de fragmentos que não portam a mutação genética; m) calcular a razão dos fragmentos que portam a mutação genética. Em uma modalidade alternativa, um método é fornecido para determinar a razão de produtos de ligação que portam um fragmento com uma mutação genética de uma população suspeita de ser heteróloga que compreende as etapas a) a h) de qualquer um dos métodos conforme descrito acima que compreende, ainda, as etapas de: i) identificar os fragmentos da etapa b); j) identificar a presença ou ausência de uma mutação genética nos fragmentos; k) identificar os produtos de ligação da etapa f) que portam os fragmentos com ou sem a mutação genética; l) determinar o número de produtos de ligação que portam os fragmentos com a mutação genética; m) determinar o número de produtos de ligação que portam os fragmentos sem a mutação genética; n) calcular a razão dos produtos de ligação que portam a mutação genética.
Nos métodos dessas modalidades, a presença ou ausência de uma mutação genética pode ser identificada na etapa j) ao alinhar a uma sequência de referência e/ou ao comparar sequências de fragmento de DNA de uma pluralidade de amostras.
Nos métodos de acordo com a invenção, uma mutação genética identificada pode ser um SNP, polimorfismo de nucleotídeo único, uma inserção, uma inversão e/ou uma translocação. Caso uma deleção e/ou inserção seja observada, o número de fragmentos e/ou produtos de ligação de uma amostra que portam a deleção e/ou inserção pode ser comparado a uma amostra de referência para identificar a deleção e/ou inserção. Uma deleção, inserção, inversão e/ou translocação também pode ser identificada com base na presença de pontos de ruptura cromossomais em fragmentos analisados.
Em outra modalidade, nos métodos conforme descrito acima, a presença ou ausência de nucleotídeos metilados é determinada nos fragmentos de DNA, fragmentos de DNA ligados, e/ou regiões genômicas de interesse. Por exemplo, o DNA da etapa a) a f) pode ser tratado com bissulfito. O tratamento de DNA com bissulfito converte resíduos de citosina em uracila, mas deixa resíduos de 5-metilcitosina não afetados. Portanto, o tratamento de bissulfito introduz modificações específicas na sequência de DNA que depende da situação de metilação de resíduos de citosina individuais, rendendo informações de resolução de nucleotídeo único sobre a situação da metilação de um segmento de DNA. Ao dividir as amostras em subamostras, em que uma das amostras é tratada e a outra não é, nucleotídeos metilados podem ser identificados. Alternativamente, sequências de uma pluralidade de amostras tratadas com bissulfito também podem ser alinhados, ou uma sequência de uma amostra tratada com bissulfito pode ser alinhada a uma sequência de referência.
Ao analisar leituras de sequência (curtas), pode ser de interesse evitar o sequenciamento dos iniciadores usados. Portanto, em um método alternativo, a sequência iniciadora pode ser removida anteriormente a etapa de sequenciamento de alto rendimento. Portanto, em uma modalidade alternativa, o método a seguir é fornecido para determinar a sequência de uma região genômica de interesse que compreende uma sequência de nucleotídeo alvo, que compreende as etapas de: a) fornecer uma amostra de DNA reticulado; b) fragmentar o DNA reticulado; c) ligar DNA reticulado fragmentado; d) reverter a reticuiação; e) opcionalmente fragmentar o DNA da etapa d), de preferência com uma enzima de restrição; f) opcionalmente ligar o DNA fragmentado da etapa d) ou e) a pelo menos um Adaptador; g) ampliar os fragmentos de DNA ligados da etapa d) ou e) que compreendem a sequência de nucleotídeo alvo com o uso de pelo menos um iniciador que, de preferência, contém uma cadeia secundária 5' que porta um sítio de reconhecimento de enzima de restrição do tipo III e (2) hibridiza na sequência de nucleotídeo alvo, ou amplia os fragmentos de DNA ligados da etapa f) com o uso de pelo menos um iniciador que (1) contém, de preferência, uma cadeia secundária 5* que porta um sítio de reconhecimento de enzima de restrição do tipo III e (2) hibridiza na sequência de nucleotídeo alvo e pelo menos um iniciador que hibridiza em pelo menos um adaptador; h) digerir as sequências de interesse de nucleotídeo ampliadas com uma enzima de restrição do tipo III, seguido por uma etapa de seleção de tamanho para remover as sequências iniciadoras de filamento duplo liberadas; i) fragmentar o DNA, de preferência por sonicação, j) ligar, opcionalmente, sequências de adaptador de filamento duplo necessárias para o sequenciamento da próxima geração k) determinar pelo menos parte da sequência dos fragmentos de DNA ligados (ampliados) da etapa d), e), f) ou g) que compreendem a sequência de nucleotídeo alvo, de preferência com o uso de sequenciamento de alto rendimento; l) identificar a variação genética na, e construir um contíguo de, região genômica de interesse a partir das sequências determinadas.
Em uma modalidade alternativa, em qualquer um dos métodos, conforme descrito no presente documento, na etapa g) iniciadores são usados portanto uma porção química, por exemplo, biotina, para a purificação ótima de fragmentos de DNA ligados (ampliados) através da ligação a um suporte sólido.
Em uma modalidade, os fragmentos de DNA ligados que compreendem a sequência de nucleotídeo alvo podem ser capturados com uma sonda de hibridização (ou sonda de captura) que hibridiza em uma sequência de nucleotídeo alvo. A sonda de hibridização pode ser fixada diretamente a um suporte sólido ou pode compreender uma porção química, por exemplo, biotina, para permitir a ligação a um suporte sólido adequado para a captura de porções químicas de biotina (por exemplo, microesferas revestidas com estreptavidina). De qualquer modo, os fragmentos de DNA ligados que compreendem uma sequência de nucleotídeo alvo são capturados permitindo, portanto, a separação de fragmentos de DNA ligados que compreendem a sequência de nucleotídeo alvo de fragmentos de DNA ligados que não compreendem a sequência de nucleotídeo alvo. Portanto, tais etapas de captura permitem o enriquecimento para fragmentos de DNA ligados que compreendem a sequencia de nucleotídeo alvo. Portanto, nos pontos, na presente invenção, nos quais uma etapa de ampliação é realizada, a qual também é uma etapa de enriquecimento, alternativamente, uma etapa de captura com uma sonda direcionada a uma sequência de nucleotídeo alvo pode ser realizada. Para uma região genômica de interesse, pelo menos uma sonda de captura para uma sequência de nucleotídeo alvo pode ser usada para a captura. Para uma região genômica de interesse, mais que uma sonda pode ser usada para múltiplas sequências de nucleotídeo alvo. Por exemplo, similar o que é descrito para o gene BRCA1, um iniciador de uma das 5 sequências de nucleotídeo alvo pode ser usado como uma sonda de captura (A, B, C, D ou E). Alternativamente, os 5 iniciadores podem ser usados de maneira combinada (A, B, C, De E), capturando a região genômica de interesse.
Em uma modalidade, uma etapa de ampliação e uma etapa de captura podem ser combinadas, por exemplo, ao primeiramente realizar uma etapa de captura e, então, uma etapa de ampliação ou vice versa.
Em uma modalidade, uma sonda de captura pode ser usada, a qual hibridiza em uma sequência adaptadora compreendida nos fragmentos de DNA ligados (ampliados).
Exemplo Este é um exemplo de uma abordagem de sequenciamento de gene integral, de acordo com a invenção, que foi usada para determinar a sequência de gene Brcal completa. As células que foram usadas eram células SUM149PT, uma linhagem celular aderente de câncer de mama, com uma deleção de um T na posição 2288 no locus Brcal (Elstrodt et al. Cancer Res, 2006). A Figura 1 mostra um esquema do método.
Cultura celular Células SUM149PT são cultivadas em pratos de 150 cm2 até placas cheias com RPMI/10% de FCS/penstrep. A divisão e contagem anteriores de um prato mostrou que um prato de 150 cm2 cheio contém -20x106 üéiuicib SUM Í49PT. Fixação e Use celular Células cultivadas são lavadas com PBS e fixadas com PBS/10% de FCS/2% de formaldeído por 10 minutos à TA. As células são subsequentemente lavadas e coletadas e absorvidas no tampão de lise. (50 mM de Tris-HCI pH 7,5, 150mM de NaCI, 5mM de EDTA, 0,5% de NP-40,1% deTX-100 e 1X) Completar inibidores de protease (Roche n° 11245200) e incubar por 10 minutos em gelo. As células são subsequentemente lavadas e absorvidas em MilliQ. Fragmentação 1: digestão As células lisadas fixadas são digeridas com Nlalll (New England Biolabs n° R0125). Ligação 1 A enzima Nlalll é inativada por calor e, subsequentemente, uma etapa de ligação é realizada com o uso de T4 DNA Ligase (Roche, n° 799009). Reticulação de reversão À amostra, Prot K (10 mg/ml) é adicionado e incubado a 65°C. RNase A (10 mg/ml, Roche n° 10109169001) é subsequentemente adicionada e a amostra é incubada a 37°C. A seguir, a extração de fenol-clorofórmio é realizada e o sobrenadante que compreende o DNA é precipitado e peletado. O pélete é dissolvido em 10 mM de Tris-HCI pH 7,5. Fragmentação 2: Segunda Digestão A amostra digerida e ligada é digerida com Nspl (New England Biolabs n° R0602S). Ligação 2: Segunda Ligação e Purificação À amostra, Prot K (10 mg/ml) é adicionado e incubado a 65°C. RNase A (10 mg/ml, Roche n° 10109169001 ) é subsequentemente adicionada e a amostra é incubada a 37°C. A seguir, a extração de fenol-clorofórmio é realizada e o sobrenadante que compreende o DNA é precipitado e peletado. O péiete é dissoivioo em tu mM de Tris-HCI pH 7,5. O modelo de enriquecimento é agora terminado e pode ser armazenado ou continuado diretamente. 5 Ampliação de fragmentos de DNA ligados: PCR
Os iniciadores usados para o enriquecimento por PCR do locus de Brcal são projetados como iniciadores únicos invertidos próximos (<50 bp) dos sítios de restrição de um fragmento de restrição de Nlalll com um espaçamento dos conjuntos de iniciador, isto é, 'pontos de vista’, de 10 aproximadamente 20 kb (consulte a Figura 2 e Tabela 1). Tabela 1. Visão geral das sequências iniciadoras usadas. Os iniciadores são nomeados (nome) com referência ao gene BRCA1, a posição no mapa de sequência (por exemplo, 50,1 (kb)) e sendo um iniciador direto (fw) ou reverso (rev), vp indica os pontos de vista, ID indica a SEQ ID No., isto é, 15 SEQ ID Nos. 1 a 10. A sequência do gene BRCA1 ao qual o iniciador corresponde também é indicada (início(5‘) e final(3')), favor notar que os iniciadores estão em uma orientação externa, isto é inversa, eles não podem formar um amplicão com o uso de DNA normal como modelo. uma reação PCR de enriquecimento consiste em 25 μl: - 2,5 μl de tampão 3 de PCR 10X (fornecido com a Polimerase de Modelo Longo de Expansão) - 0,5 μl de dNTP (10 mM) - 0,5 μl de iniciador direto (de uma diluição de 1/7 a partir de uma matéria prima iniciadora de 1 μg /μl) - 0,5 μl de iniciador reverso (de uma diluição de 1/7 a partir de uma matéria prima iniciadora de 1 μg/μl) - 0,375 μl de Polimerase de Modelo Longo de Expansão (Roche n° 11759060001) - 100 ng de modelo de enriquecimento - X μl de Milli-Q para um volume total de 25 μl
Sequenciamento dos fragmentos de DNA ligados ampliados Prossiga com a preparação de biblioteca para o sequenciamento SOLiD, de acordo com os protocolos padrão de SOLiD.
Resultados A distribuição de leitura dos diferentes pontos de vista foi mais alta acerca do sítio do ponto de vista. Estatísticas adicionais são mostradas na Tabela 2. Leituras de sequência das bibliotecas de ponto de vista C, D e E identificaram a mutação 2288delT. Também foram determinadas quais sequências do gene BRCA1 não foram cobertas, do ponto de vista A, 15.807 pares de base não foram cobertos, do B, 50.124 b. De C, D e E todas as BRCA1 sequências foram cobertas. Tabela 2. Estatísticas de leitura de sequência por ponto de vista. As estatísticas de leitura de sequência por biblioteca de ponto de vista (vp, A-E) são indicadas. ívi (ieituras compatíveis com BRCA1 ), TR (número total de leituras), % de MtT (% das leituras totais compatíveis com alvo), média 5 (cobertura média), mediano (cobertura de mediano), % de nt 20x (a % de nucleotídeos do gene BRCA1 que foram cobertos mais de 20 vezes). Portanto, dos pontos de vista únicos C, D e E, o BRCA1 completo de 100 kb foi coberto, do ponto de vista A, 85 kb do gene BRCA1 gene foram cobertos, do ponto de vista B, 50 kb do gene BRCA1 foram cobertos e dos 10 pontos de vista C, D e E, a mutação 2288delT foi confirmada.
Claims (29)
1. Método para construir um contíguo de uma região genômica de interesse que compreende uma sequência de nucleotídeo alvo CARACTERIZADO por compreender fragmentar um DNA reticulado, ligar o DNA reticulado fragmentado, reverter a reticulação e determinar pelo menos parte das sequências de fragmentos de DNA ligados que compreendem a sequência de nucleotídeo alvo e usar as sequências determinadas para construir um contíguo da região genômica de interesse.
2. Método para construir um contíguo de uma região genômica de interesse que compreende uma sequência de nucleotídeo alvo, de acordo com a reivindicação 1, CARACTERIZADO por compreender as etapas de: a) fornecer uma amostra de DNA reticulado; b) fragmentar o DNA reticulado; c) ligar o DNA reticulado fragmentado; d) reverter a reticulação; e) fragmentar, opcionalmente, o DNA da etapa d), de preferência com uma enzima de restrição; f) ligar, opcionalmente, o DNA fragmentado da etapa d) ou e) para pelo menos um adaptador; g) opcional e preferencialmente ampliar os fragmentos de DNA ligados da etapa d) ou e) que compreendem a sequência de nucleotídeo alvo com o uso de pelo menos um iniciador que hibridiza nas sequência de nucleotídeo alvo ou ampliar os fragmentos de DNA ligados da etapa f) com o uso de pelo menos um iniciador que hibridiza na sequência de nucleotídeo alvo e pelo menos um iniciador que hibridiza em pelo menos um adaptador; h) determinar pelo menos parte da sequência dos fragmentos de DNA ligados (ampliados) da etapa d), e), f) ou g) que compreendem a sequência de nucleotídeo alvo, de preferência com o uso do sequenciamento de alto rendimento; i) construir um contíguo da região genômica de interesse a partir das sequências determinadas.
3. Método para construir um contíguo de uma região genômica de interesse que compreende uma sequência de nucleotídeo alvo, de acordo com a reivindicação 1, CARACTERIZADO por compreender as etapas de: a) fornecer uma amostra de DNA reticulado; b) fragmentar o DNA reticulado; c) ligar o DNA reticulado fragmentado; d) reverter a reticulação; e) fragmentar, opcionalmente, o DNA da etapa d), de preferência com uma enzima de restrição f) circularizar o DNA da etapa d) ou e); g) opcional e preferencialmente ampliar o DNA circularizado que compreende a sequência de nucleotídeo alvo com o uso, de preferência, de pelo menos um iniciador que hibridiza na sequência de nucleotídeo alvo; h) determinar pelo menos parte da sequência dos fragmentos de DNA ligados (ampliados) que compreende o nucleotídeo alvo com o uso do sequenciamento de alto rendimento; i) construir um contíguo da região genômica de interesse a partir das sequências determinadas.
4. Método para construir um contíguo de uma região genômica de interesse, de acordo com as reivindicações 2 ou 3, CARACTERIZADO pelo fato de que a região genômica de interesse compreende, adicionalmente, uma ou mais sequências de nucleotídeo alvo e em que, na etapa de ampliação (g), um iniciador é fornecido, o qual hibridiza na sequência de nucleotídeo alvo, e um ou mais iniciadores são fornecidos para os um ou mais nucleotídeos alvo adicionais correspondentes, em que os fragmentos de DNA ligados são ampliados, ou o DNA circularizado é ampliado, com o uso de iniciadores.
5. Método, de acordo com qualquer uma das reivindicações de 2 a 4, CARACTERIZADO pelo fato de que a etapa de fragmentação b) compreende sonicação, seguida pelo DNA enzimático e reparo.
6. Método, de acordo com qualquer uma das reivindicações de 2 a 4, CARACTERIZADO pelo fato de que a etapa de fragmentação b) compreende fragmentar com uma enzima de restrição.
7. Método, de acordo com a reivindicação 5 ou 6, CARACTERIZADO pelo fato de que a etapa de ligação c) é realizada na presença de um adaptador, ligando as sequências de adaptador entre os fragmentos.
8. Método, de acordo com as reivindicações 6 ou 7, CARACTERIZADO pelo fato de que, na etapa b), uma pluralidade de subamostras é processada e, para cada subamostra, enzimas de restrição com sítios de reconhecimento diferentes são usados.
9. Método, de acordo com a reivindicação 8, CARACTERIZADO pelo fato de que a etapa de fragmentação e) compreende uma enzima de restrição com uma sequência de reconhecimento que é mais longa que a sequência de reconhecimento da enzima de restrição da etapa b).
10. Método, de acordo com qualquer uma das reivindicações precedentes, CARACTERIZADO pelo fato de que que as sequências de múltiplas regiões genômicas de interesses são determinadas.
11. Método, de acordo com qualquer uma das reivindicações de 2 a 10, CARACTERIZADO pelo fato de que um identificador é incluído em pelo menos um dos iniciadores de oligonucleotídeo da etapa g).
12. Método, de acordo com qualquer uma das reivindicações de 2 a 11, CARACTERIZADO pelo fato de que anteriormente ou após a etapa de ampliação g), uma etapa de seleção de tamanho é realizada.
13. Método, de acordo com a reivindicação 12, CARACTERIZADO pelo fato de que a etapa de seleção de tamanho é realizada com o uso de cromatografia de extração em gel, eletroforese em gel ou centrifugação de gradiente de densidade.
14. Método, de acordo com qualquer uma das reivindicações de 12 a 13, CARACTERIZADO pelo fato de que o DNA é selecionado de um tamanho entre 20 e 20.0000 pares de base, de preferência 50 e 10.0000 pares de base, com máxima preferência entre 100 e 3.000 pares de base.
15. Método, de acordo com qualquer uma das reivindicações de 1 a 14, CARACTERIZADO pelo fato de que, caso a ploidia em uma célula de uma região genômica de interesse seja maior que 1, para cada ploidia um contíguo é construído.
16. Método, de acordo com qualquer uma das reivindicações de 2 a 15, CARACTERIZADO pelo fato de que a etapa i) de construção de um contíguo compreende as etapas de: 1) identificar os fragmentos da etapa b); 2) designar os fragmentos a uma região genômica; 3) construir um contíguo para a região genômica.
17. Método, de acordo com a reivindicação 16, CARACTERIZADO pelo fato de que a etapa 2) de designação dos fragmentos a uma região genômica compreende identificar os diferentes produtos de ligação da etapa f) e acoplar os diferentes produtos de ligação aos fragmentos identificados.
18. Método para identificar a presença ou ausência de uma mutação genética CARACTERIZADO por compreender as etapas a) a i), conforme definido em qualquer uma das reivindicações de 2 a 17, em que contíguos são construídos para uma pluralidade de amostras, que compreende, ainda, as etapas de: j) alinhar os contíguos de uma pluralidade de amostras; k) identificar a presença ou ausência de uma mutação genética nas regiões genômicas de interesse a partir da pluralidade de amostras.
19. Método para identificar a presença ou ausência de uma mutação genética CARACTERIZADO por compreender as etapas a) a i), conforme definido em qualquer uma das reivindicações de 2 a 17, que compreende as etapas adicionais de: j) alinhar o contíguo a uma sequência de referência. k) identificar a presença ou ausência de uma mutação genética na região genômica de interesse.
20. Método para identificar a presença ou ausência de uma mutação genética CARACTERIZADO por compreender as etapas a) a h), conforme definidas em qualquer uma das reivindicações de 2 a 14, que compreende as etapas adicionais: i) alinhar as sequências determinadas dos fragmentos de DNA ligados (ampliados) a uma sequência de referência. j) identificar a presença ou ausência de uma mutação genética nas sequências determinadas.
21. Método para identificar a presença ou ausência de uma mutação genética CARACTERIZADO por compreender as etapas a) a h), conforme definido em qualquer uma das reivindicações de 2 a 14, em que uma pluralidade de amostras sequências de fragmentos de DNA ligados (ampliados) é determinada, que compreende, ainda, as etapas de: i) alinhar as sequências determinadas dos fragmentos de DNA ligados (ampliados) de uma pluralidade de amostras. j) identificar a presença ou ausência de uma mutação genética nas sequências determinadas.
22. Método, de acordo com qualquer uma das reivindicações de 2 a 21, CARACTERIZADO pelo fato de que fragmentos de DNA ligados (amplificados) compreendendo a sequência de nucleotídeos alvo são capturados com uma sonda de captura para separar fragmentos de DNA ligados (amplificados) compreendendo a sequência de nucleotídeos alvo de fragmentos de DNA ligados (amplificados) que não compreendem a sequência de nucleotídeos alvo.
23. Método para determinar a razão de fragmentos que portam uma mutação genética de uma população de células suspeita de ser heteróloga CARACTERIZADO por compreender as etapas a) a h), conforme definidas em qualquer uma das reivindicações de 2 a 14, que compreende as etapas adicionais de: i) identificar os fragmentos da etapa b); j) identificar a presença ou ausência de uma mutação genética nos fragmentos; k) determinar o número de fragmentos que portam a mutação genética; l) determinar o número de fragmentos que não portam a mutação genética; m) calcular a razão dos fragmentos que portam a mutação genética.
24. Método para determinar a razão dos produtos de ligação que portam um fragmento com uma mutação genética de uma população de células suspeita de ser heteróloga CARACTERIZADO por compreender as etapas a) a h), conforme definidas em qualquer uma das reivindicações de 2 a 14, que compreende, ainda, as etapas de: i) identificar os fragmentos da etapa b); j) identificar a presença ou ausência de uma mutação genética nos fragmentos; k) identificar os fragmentos de DNA ligados que portam os fragmentos com ou sem a mutação genética; l) determinar o número dos fragmentos de DNA ligados que portam os fragmentos com a mutação genética; m) determinar o número de produtos de ligação que portam os fragmentos sem a mutação genética; n) calcular a razão de produtos de ligação que portam a mutação genética.
25. Método, de acordo com qualquer uma das reivindicações de 23 a 24, CARACTERIZADO pelo fato de que, na etapa j), a presença ou ausência de uma mutação genética é identificada pelo alinhamento a uma sequência de referência e/ou ao comparar fragmento sequências de uma pluralidade de amostras.
26. Método, de acordo com qualquer uma das reivindicações de 18 a 25, CARACTERIZADO pelo fato de que uma mutação genética é um SNP, uma deleção, uma inserção, uma inversão e/ou uma translocação.
27. Método, de acordo com a reivindicação 26, CARACTERIZADO pelo fato de que uma deleção e/ou inserção é identificada ao comparar o número de fragmentos e/ou produtos de ligação a partir de uma amostra que porta a deleção e/ou inserção com uma amostra de referência.
28. Método, de acordo com a reivindicação 26, CARACTERIZADO pelo fato de que uma deleção, inserção, inversão e/ou translocação é identificada com base na presença de pontos de ruptura cromossomais em fragmentos analisados.
29. Método, de acordo com qualquer uma das reivindicações precedentes, CARACTERIZADO pelo fato de que a presença ou ausência de nucleotídeos metilados é determinada em fragmentos de DNA, fragmentos de DNA ligados, e/ou regiões genômicas de interesse.
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US36277810P | 2010-07-09 | 2010-07-09 | |
| US61/362,778 | 2010-07-09 | ||
| PCT/NL2011/050504 WO2012005595A2 (en) | 2010-07-09 | 2011-07-08 | V3-d genomic region of interest sequencing strategies |
Publications (3)
| Publication Number | Publication Date |
|---|---|
| BR112013000552A2 BR112013000552A2 (pt) | 2016-05-24 |
| BR112013000552B1 true BR112013000552B1 (pt) | 2021-03-02 |
| BR112013000552B8 BR112013000552B8 (pt) | 2023-01-17 |
Family
ID=44629077
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| BR112013000552A BR112013000552B8 (pt) | 2010-07-09 | 2011-07-08 | Estratégias de sequenciamento de região de interesse genômica em 3d |
Country Status (26)
| Country | Link |
|---|---|
| US (2) | US12006538B2 (pt) |
| EP (3) | EP2591125B1 (pt) |
| JP (1) | JP5977234B2 (pt) |
| KR (1) | KR101862756B1 (pt) |
| CN (1) | CN103180459B (pt) |
| AU (1) | AU2011274642B2 (pt) |
| BR (1) | BR112013000552B8 (pt) |
| CA (1) | CA2804450C (pt) |
| CY (1) | CY1121702T1 (pt) |
| DK (2) | DK3360975T3 (pt) |
| ES (2) | ES2667346T3 (pt) |
| HR (1) | HRP20180593T1 (pt) |
| HU (1) | HUE037359T2 (pt) |
| IL (2) | IL224127B (pt) |
| LT (1) | LT2591125T (pt) |
| NO (1) | NO2591125T3 (pt) |
| NZ (1) | NZ606228A (pt) |
| PL (2) | PL2591125T3 (pt) |
| PT (2) | PT3360975T (pt) |
| RS (1) | RS57295B1 (pt) |
| RU (1) | RU2603082C2 (pt) |
| SG (1) | SG186954A1 (pt) |
| SI (1) | SI2591125T1 (pt) |
| SM (1) | SMT201800213T1 (pt) |
| WO (1) | WO2012005595A2 (pt) |
| ZA (1) | ZA201300375B (pt) |
Families Citing this family (69)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| JP2012525147A (ja) | 2009-04-30 | 2012-10-22 | グッド スタート ジェネティクス, インコーポレイテッド | 遺伝マーカーを評価するための方法および組成物 |
| US12129514B2 (en) | 2009-04-30 | 2024-10-29 | Molecular Loop Biosolutions, Llc | Methods and compositions for evaluating genetic markers |
| CA2804450C (en) | 2010-07-09 | 2022-10-11 | Max Jan van Min | 3-d genomic region of interest sequencing strategies |
| US9163281B2 (en) | 2010-12-23 | 2015-10-20 | Good Start Genetics, Inc. | Methods for maintaining the integrity and identification of a nucleic acid template in a multiplex sequencing reaction |
| US9249460B2 (en) | 2011-09-09 | 2016-02-02 | The Board Of Trustees Of The Leland Stanford Junior University | Methods for obtaining a sequence |
| JP2014531908A (ja) | 2011-10-14 | 2014-12-04 | プレジデント アンド フェローズ オブ ハーバード カレッジ | 構造アッセンブリによる配列決定 |
| CA2852665A1 (en) | 2011-10-17 | 2013-04-25 | Good Start Genetics, Inc. | Analysis methods |
| ES2991004T3 (es) | 2011-12-22 | 2024-12-02 | Harvard College | Métodos para la detección de analitos |
| ES2952728T3 (es) | 2011-12-22 | 2023-11-03 | Harvard College | Métodos para la detección de analitos |
| US8209130B1 (en) | 2012-04-04 | 2012-06-26 | Good Start Genetics, Inc. | Sequence assembly |
| US10227635B2 (en) | 2012-04-16 | 2019-03-12 | Molecular Loop Biosolutions, Llc | Capture reactions |
| WO2013184754A2 (en) | 2012-06-05 | 2013-12-12 | President And Fellows Of Harvard College | Spatial sequencing of nucleic acids using dna origami probes |
| US9411930B2 (en) | 2013-02-01 | 2016-08-09 | The Regents Of The University Of California | Methods for genome assembly and haplotype phasing |
| CA3209385A1 (en) * | 2013-02-01 | 2014-08-07 | The Regents Of The University Of California | Methods for genome assembly and haplotype phasing |
| EP2959011A1 (en) * | 2013-02-19 | 2015-12-30 | Cergentis B.V. | Sequencing strategies for genomic regions of interest |
| US10138509B2 (en) | 2013-03-12 | 2018-11-27 | President And Fellows Of Harvard College | Method for generating a three-dimensional nucleic acid containing matrix |
| WO2014152421A1 (en) | 2013-03-14 | 2014-09-25 | Good Start Genetics, Inc. | Methods for analyzing nucleic acids |
| EP2971278B1 (en) * | 2013-03-15 | 2022-08-10 | The Broad Institute, Inc. | Methods for determining multiple interactions between nucleic acids in a cell |
| US10287621B2 (en) | 2013-04-11 | 2019-05-14 | Pelin Sahlén | Targeted chromosome conformation capture |
| RU2690935C2 (ru) | 2013-06-04 | 2019-06-06 | Президент Энд Фэллоуз Оф Харвард Коллидж | Направляемая рнк регуляция транскрипции |
| CN111705116A (zh) * | 2013-07-19 | 2020-09-25 | 路德维格癌症研究有限公司 | 全基因组且靶向的单体型重构 |
| GB2517936B (en) * | 2013-09-05 | 2016-10-19 | Babraham Inst | Chromosome conformation capture method including selection and enrichment steps |
| US11694764B2 (en) | 2013-09-27 | 2023-07-04 | University Of Washington | Method for large scale scaffolding of genome assemblies |
| US10851414B2 (en) | 2013-10-18 | 2020-12-01 | Good Start Genetics, Inc. | Methods for determining carrier status |
| GB201320351D0 (en) * | 2013-11-18 | 2014-01-01 | Erasmus Universiteit Medisch Ct | Method |
| US11053548B2 (en) | 2014-05-12 | 2021-07-06 | Good Start Genetics, Inc. | Methods for detecting aneuploidy |
| EP2960818A1 (en) * | 2014-06-24 | 2015-12-30 | Institut Pasteur | Method, device, and computer program for assembling pieces of chromosomes from one or several organisms |
| WO2016007839A1 (en) | 2014-07-11 | 2016-01-14 | President And Fellows Of Harvard College | Methods for high-throughput labelling and detection of biological features in situ using microscopy |
| WO2016019360A1 (en) | 2014-08-01 | 2016-02-04 | Dovetail Genomics Llc | Tagging nucleic acids for sequence assembly |
| US20160048608A1 (en) | 2014-08-15 | 2016-02-18 | Good Start Genetics, Inc. | Systems and methods for genetic analysis |
| WO2016040446A1 (en) | 2014-09-10 | 2016-03-17 | Good Start Genetics, Inc. | Methods for selectively suppressing non-target sequences |
| CA2999708A1 (en) | 2014-09-24 | 2016-03-31 | Good Start Genetics, Inc. | Process control for increased robustness of genetic assays |
| WO2016089920A1 (en) * | 2014-12-01 | 2016-06-09 | The Broad Institute, Inc. | Method for in situ determination of nucleic acid proximity |
| US10066259B2 (en) * | 2015-01-06 | 2018-09-04 | Good Start Genetics, Inc. | Screening for structural variants |
| EP4030437A1 (en) | 2015-02-17 | 2022-07-20 | Dovetail Genomics, LLC | Nucleic acid sequence assembly |
| GB2554572B (en) | 2015-03-26 | 2021-06-23 | Dovetail Genomics Llc | Physical linkage preservation in DNA storage |
| US10395759B2 (en) * | 2015-05-18 | 2019-08-27 | Regeneron Pharmaceuticals, Inc. | Methods and systems for copy number variant detection |
| EP3317420B1 (en) | 2015-07-02 | 2021-10-20 | Arima Genomics, Inc. | Accurate molecular deconvolution of mixtures samples |
| US20180282799A1 (en) * | 2015-10-05 | 2018-10-04 | Koninklijke Nederlandse Akademie Van Wetenschappen | Targeted locus amplification using cloning strategies |
| WO2017066908A1 (zh) | 2015-10-19 | 2017-04-27 | 安诺优达基因科技(北京)有限公司 | 构建高分辨率、大信息量单细胞Hi-C文库的方法 |
| EP3365445B1 (en) | 2015-10-19 | 2023-05-31 | Dovetail Genomics, LLC | Methods for genome assembly, haplotype phasing, and target independent nucleic acid detection |
| GB201518843D0 (en) | 2015-10-23 | 2015-12-09 | Isis Innovation | Method of analysing DNA sequences |
| JP6882282B2 (ja) | 2015-11-03 | 2021-06-02 | プレジデント アンド フェローズ オブ ハーバード カレッジ | 三次元核酸含有マトリックスの立体撮像のための方法と装置 |
| CN115369161A (zh) | 2015-12-04 | 2022-11-22 | 10X 基因组学有限公司 | 用于核酸分析的方法和组合物 |
| US10975417B2 (en) * | 2016-02-23 | 2021-04-13 | Dovetail Genomics, Llc | Generation of phased read-sets for genome assembly and haplotype phasing |
| EP4613756A3 (en) | 2016-04-25 | 2025-11-12 | President And Fellows Of Harvard College | Hybridization chain reaction methods for in situ molecular detection |
| GB201608000D0 (en) * | 2016-05-06 | 2016-06-22 | Oxford Biodynamics Ltd | Chromosome detection |
| CN105839196B (zh) * | 2016-05-11 | 2018-04-17 | 北京百迈客生物科技有限公司 | 一种真核生物DNA的Hi‑C高通量测序建库方法 |
| EP3455356B1 (en) | 2016-05-13 | 2021-08-04 | Dovetail Genomics LLC | Recovering long-range linkage information from preserved samples |
| KR101915701B1 (ko) * | 2016-06-16 | 2018-11-07 | 한국한의학연구원 | 돌연변이 발생률의 측정 방법 |
| EP3507364A4 (en) | 2016-08-31 | 2020-05-20 | President and Fellows of Harvard College | METHODS OF GENERATING NUCLEIC ACID SEQUENCE LIBRARIES FOR IN SITU FLUORESCENT SEQUENCING DETECTION |
| CN109923216B (zh) | 2016-08-31 | 2024-08-02 | 哈佛学院董事及会员团体 | 将生物分子的检测组合到使用荧光原位测序的单个试验的方法 |
| CA3045070A1 (en) | 2016-12-01 | 2018-06-07 | Oxford Biodynamics Limited | Application of epigenetic chromsomal interactions in cancer diagnostics |
| US11185568B2 (en) | 2017-04-14 | 2021-11-30 | President And Fellows Of Harvard College | Methods for generation of cell-derived microfilament network |
| CN107119120A (zh) * | 2017-05-04 | 2017-09-01 | 河海大学常州校区 | 一种基于染色质3d构象技术的关键作用分子检测方法 |
| WO2019104034A1 (en) * | 2017-11-21 | 2019-05-31 | Arima Genomics, Inc. | Preserving spatial-proximal contiguity and molecular contiguity in nucleic acid templates |
| US12378592B2 (en) | 2018-01-31 | 2025-08-05 | Dovetail Genomics, Llc. | Sample prep for DNA linkage recovery |
| KR20250162921A (ko) | 2018-04-09 | 2025-11-19 | 더 보드 오브 트러스티스 오브 더 리랜드 스탠포드 쥬니어 유니버시티 | 계내 유전자 시퀀싱의 방법 |
| CN112770776B (zh) | 2018-07-30 | 2025-08-19 | 瑞德库尔有限责任公司 | 用于样品处理或分析的方法和系统 |
| WO2020076976A1 (en) | 2018-10-10 | 2020-04-16 | Readcoor, Inc. | Three-dimensional spatial molecular indexing |
| US20220251640A1 (en) * | 2019-05-17 | 2022-08-11 | The Broad Institute, Inc. | Methods of determination of genome architecture and epigenetic profile |
| GB202006546D0 (en) | 2020-05-04 | 2020-06-17 | Cergentis B V | Method |
| WO2021224225A1 (en) | 2020-05-04 | 2021-11-11 | Cergentis B.V. | Method |
| GB202006550D0 (en) | 2020-05-04 | 2020-06-17 | Cergentis B V | Method |
| WO2021224233A1 (en) | 2020-05-04 | 2021-11-11 | Cergentis B.V. | Method |
| US20220049303A1 (en) | 2020-08-17 | 2022-02-17 | Readcoor, Llc | Methods and systems for spatial mapping of genetic variants |
| EP4251772A1 (en) | 2020-11-26 | 2023-10-04 | Stichting Het Nederlands Kanker Instituut- Antoni van Leeuwenhoek Ziekenhuis | Personalized tumor markers |
| US20240263228A1 (en) * | 2021-05-21 | 2024-08-08 | The Board Of Trustees Of The Leland Stanford Junior University | Next-Generation Volumetric in Situ Sequencing |
| GB202111194D0 (en) | 2021-08-03 | 2021-09-15 | Cergentis B V | Method |
Family Cites Families (69)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US5077204A (en) * | 1984-06-21 | 1991-12-31 | Chiron Corporation | Yeast endopeptidase for basic amino-acid site cleavage, preparation and use |
| US9328391B1 (en) | 1984-08-22 | 2016-05-03 | The United States Of America As Represented By The Secretary, Department Of Health And Human Services | Cloning and expression of HIV-1 DNA |
| JPS6226300A (ja) | 1984-10-10 | 1987-02-04 | セントコ−・インコ−ポレ−テツド | Htlv−3dnaのクロ−ニングおよび発現 |
| US5221518A (en) * | 1984-12-14 | 1993-06-22 | Mills Randell L | DNA sequencing apparatus |
| US5258507A (en) * | 1990-11-08 | 1993-11-02 | Amoco Corporation | Labeling reagents useful for the chemical attachment of nitrophenyl derivative ligands to DNA probes |
| US6060644A (en) * | 1994-03-24 | 2000-05-09 | Iowa State University Research Foundation, Inc. | Isolation and use of cuticular lipid genes |
| EP2369007B1 (en) * | 1996-05-29 | 2015-07-29 | Cornell Research Foundation, Inc. | Detection of nucleic acid sequence differences using coupled ligase detection and polymerase chain reactions |
| JP2002500623A (ja) * | 1996-11-07 | 2002-01-08 | ザ ボード オブ トラスティーズ オブ ザ リーランド スタンフォード ジュニア ユニヴァーシティー | スプルーティタンパク質およびコーディング配列 |
| US6261804B1 (en) * | 1997-01-21 | 2001-07-17 | The General Hospital Corporation | Selection of proteins using RNA-protein fusions |
| ATE273381T1 (de) * | 1997-02-12 | 2004-08-15 | Eugene Y Chan | Verfahren zur analyse von polymeren |
| AU1463299A (en) * | 1997-12-02 | 1999-06-16 | E.I. Du Pont De Nemours And Company | Branched chain amino acid biosynthetic enzymes |
| US6455692B1 (en) * | 1998-08-04 | 2002-09-24 | Transgenomic, Inc. | Method of concentrating polynucleotides using MIPC |
| US6291168B1 (en) * | 1998-10-27 | 2001-09-18 | Auburn University | Nucleic acid sequences diagnostic for pathogenic E.coli O157:H7, methods of identification and kit therefore |
| US20050232900A1 (en) * | 1999-05-18 | 2005-10-20 | Crucell Holland B.V. | Serotype of adenovirus and uses thereof |
| AU1824101A (en) * | 1999-06-18 | 2001-01-09 | Elitra Pharmaceuticals, Inc. | Nucleotide sequences of moraxella catarrhalis genome |
| US7470507B2 (en) * | 1999-09-01 | 2008-12-30 | Whitehead Institute For Biomedical Research | Genome-wide location and function of DNA binding proteins |
| US20040014195A1 (en) * | 1999-12-29 | 2004-01-22 | Diversa Corporation | Nitrilases, nucleic acids encoding them and methods for making and using them |
| US20060292560A1 (en) * | 2000-08-14 | 2006-12-28 | Robert Burgess | Transcription factor target gene discovery |
| US6709861B2 (en) * | 2000-11-17 | 2004-03-23 | Lucigen Corp. | Cloning vectors and vector components |
| US20030054347A1 (en) * | 2001-04-27 | 2003-03-20 | The Regents Of The University Of Michigan | Latrophilin Polynucleotides |
| US20030013863A1 (en) * | 2001-07-16 | 2003-01-16 | Buchholz Wallace G. | High resolution DNA size standards |
| US20030157705A1 (en) * | 2001-08-10 | 2003-08-21 | Fodor William L. | Engineered recombinant molecule that regulates humoral and cellular effector functions of the immune system |
| AUPR823601A0 (en) * | 2001-10-12 | 2001-11-08 | University Of Queensland, The | Automated genotyping |
| WO2003102147A2 (en) * | 2002-05-29 | 2003-12-11 | University Of Washington | Mutants of gad65 and ian5 relating to diabetes |
| AU2003252591A1 (en) * | 2002-08-30 | 2004-03-29 | Japan Science And Technology Corporation | Method of targeted gene disruption, genome of hyperthermostable bacterium and genome chip using the same |
| CA2500129C (en) * | 2002-09-30 | 2011-03-29 | F. Hoffmann-La Roche Ag | Oligonucleotides for genotyping thymidylate synthase gene |
| US7575865B2 (en) * | 2003-01-29 | 2009-08-18 | 454 Life Sciences Corporation | Methods of amplifying and sequencing nucleic acids |
| JP2006519605A (ja) * | 2003-03-06 | 2006-08-31 | チルドレンズ ホスピタル, インコーポレイテッド | 類型不能haemophilusinfluenzaeの中耳炎単離株の遺伝子 |
| FR2852317B1 (fr) * | 2003-03-13 | 2006-08-04 | Biopuces a sondes et leurs methodes d'utilisation | |
| WO2005026203A2 (en) * | 2003-09-18 | 2005-03-24 | The Government Of The United States Of America, As Represented By The Secretary, Department Of Health And Human Services | Dna promoters and anthrax vaccines |
| US20060150283A1 (en) * | 2004-02-13 | 2006-07-06 | Nickolai Alexandrov | Sequence-determined DNA fragments and corresponding polypeptides encoded thereby |
| US20060107345A1 (en) * | 2003-09-30 | 2006-05-18 | Nickolai Alexandrov | Sequence-determined DNA fragments and corresponding polypeptides encoded thereby |
| US20050221341A1 (en) * | 2003-10-22 | 2005-10-06 | Shimkets Richard A | Sequence-based karyotyping |
| EP1694869A2 (en) * | 2003-11-10 | 2006-08-30 | Investigen, Inc. | Methods of preparing nucleic acid for detection |
| US20060073501A1 (en) * | 2004-09-10 | 2006-04-06 | Van Den Boom Dirk J | Methods for long-range sequence analysis of nucleic acids |
| US20070048748A1 (en) * | 2004-09-24 | 2007-03-01 | Li-Cor, Inc. | Mutant polymerases for sequencing and genotyping |
| US20060223073A1 (en) * | 2005-03-31 | 2006-10-05 | Boyes Barry E | Methods of using a DNase I-like enzyme |
| US7601499B2 (en) * | 2005-06-06 | 2009-10-13 | 454 Life Sciences Corporation | Paired end sequencing |
| US8407013B2 (en) * | 2005-06-07 | 2013-03-26 | Peter K. Rogan | AB initio generation of single copy genomic probes |
| EP1907571B1 (en) * | 2005-06-15 | 2017-04-26 | Complete Genomics Inc. | Nucleic acid analysis by random mixtures of non-overlapping fragments |
| CA2613248A1 (en) * | 2005-06-23 | 2006-12-28 | Keygene N.V. | Improved strategies for sequencing complex genomes using high throughput sequencing technologies |
| WO2006137733A1 (en) * | 2005-06-23 | 2006-12-28 | Keygene N.V. | Strategies for high throughput identification and detection of polymorphisms |
| NZ564262A (en) | 2005-07-04 | 2010-02-26 | Univ Erasmus Medical Ct | Chromosome conformation capture-on-chip (4C) assay |
| US20090143240A1 (en) * | 2005-08-25 | 2009-06-04 | Harbison Christopher T | Novel Methods for Genome-Wide Location Analysis |
| WO2007030678A2 (en) * | 2005-09-07 | 2007-03-15 | Whitehead Institute For Biomedical Research | Methods of genome-wide location analysis in stem cells |
| US8071310B2 (en) | 2005-11-14 | 2011-12-06 | Keygene Nv | Method for the high throughput screening of transposon tagging populations and massive parallel sequence identification of insertion sites |
| JP5285431B2 (ja) * | 2005-12-28 | 2013-09-11 | ザ ユーエイビー リサーチ ファウンデイション | 肺炎球菌血清型 |
| GB0603251D0 (en) * | 2006-02-17 | 2006-03-29 | Isis Innovation | DNA conformation |
| WO2008008845A2 (en) | 2006-07-11 | 2008-01-17 | Microchips, Inc. | Multi-reservoir pump device for dialysis, biosensing, or delivery of substances |
| WO2008015396A2 (en) * | 2006-07-31 | 2008-02-07 | Solexa Limited | Method of library preparation avoiding the formation of adaptor dimers |
| DE112007002880T5 (de) * | 2006-11-27 | 2009-10-29 | Dsm Ip Assets B.V. | Verfahren zur Herstellung von Hydroxytyrosol |
| AU2007334393A1 (en) * | 2006-12-14 | 2008-06-26 | Life Technologies Corporation | Methods and apparatus for measuring analytes using large scale FET arrays |
| AU2008204338B2 (en) | 2007-01-11 | 2014-03-06 | Erasmus University Medical Center | Circular chromosome conformation capture (4C) |
| EP2126765B1 (en) * | 2007-01-26 | 2011-08-24 | Illumina Inc. | Nucleic acid sequencing system and method |
| JPWO2008111453A1 (ja) * | 2007-03-07 | 2010-06-24 | 国立大学法人 東京大学 | Dna断片の増幅方法 |
| US7932030B2 (en) * | 2007-04-05 | 2011-04-26 | The Board Of Supervisors Of Louisiana State University And Agricultural And Mechanical College | System for pulling out regulatory elements using yeast |
| US20080248958A1 (en) * | 2007-04-05 | 2008-10-09 | Hollenbach Andrew D | System for pulling out regulatory elements in vitro |
| CA2689356A1 (en) * | 2007-06-01 | 2008-12-11 | 454 Life Sciences Corporation | System and meth0d for identification of individual samples from a multiplex mixture |
| EP2053132A1 (en) * | 2007-10-23 | 2009-04-29 | Roche Diagnostics GmbH | Enrichment and sequence analysis of geomic regions |
| US8518640B2 (en) * | 2007-10-29 | 2013-08-27 | Complete Genomics, Inc. | Nucleic acid sequencing and process |
| US8263367B2 (en) * | 2008-01-25 | 2012-09-11 | Agency For Science, Technology And Research | Nucleic acid interaction analysis |
| US8296075B2 (en) * | 2008-05-21 | 2012-10-23 | Mito Tech, LLC | Computer system and computer-facilitated method for nucleic acid sequence alignment and analysis |
| CN102203251A (zh) * | 2008-05-23 | 2011-09-28 | 生命科技公司 | 用于dna提取的方法和试剂盒 |
| FR2945545B1 (fr) * | 2009-05-14 | 2011-08-05 | Univ Aix Marseille Ii | Methode de detection d'adn procaryote extrait d'un echantillon de selles |
| RU2552215C2 (ru) * | 2009-09-01 | 2015-06-10 | Конинклейке Филипс Электроникс Н.В. | Устройство и способ отбора нуклеиновых кислот с помощью микроматриц |
| EP2354243A1 (en) | 2010-02-03 | 2011-08-10 | Lexogen GmbH | Complexity reduction method |
| JP5901046B2 (ja) * | 2010-02-19 | 2016-04-06 | 国立大学法人 千葉大学 | OATP1B3mRNAの新規な選択的スプライシングバリアント |
| CA2804450C (en) | 2010-07-09 | 2022-10-11 | Max Jan van Min | 3-d genomic region of interest sequencing strategies |
| US10858699B2 (en) * | 2016-08-30 | 2020-12-08 | Integrated Dna Technologies, Inc. | Cleavable hairpin primers |
-
2011
- 2011-07-08 CA CA2804450A patent/CA2804450C/en active Active
- 2011-07-08 SI SI201131467T patent/SI2591125T1/en unknown
- 2011-07-08 SG SG2013001029A patent/SG186954A1/en unknown
- 2011-07-08 JP JP2013518301A patent/JP5977234B2/ja active Active
- 2011-07-08 DK DK18163123.5T patent/DK3360975T3/da active
- 2011-07-08 EP EP11736190.7A patent/EP2591125B1/en active Active
- 2011-07-08 BR BR112013000552A patent/BR112013000552B8/pt active IP Right Grant
- 2011-07-08 HU HUE11736190A patent/HUE037359T2/hu unknown
- 2011-07-08 WO PCT/NL2011/050504 patent/WO2012005595A2/en not_active Ceased
- 2011-07-08 US US13/807,662 patent/US12006538B2/en active Active
- 2011-07-08 PT PT181631235T patent/PT3360975T/pt unknown
- 2011-07-08 PL PL11736190T patent/PL2591125T3/pl unknown
- 2011-07-08 LT LTEP11736190.7T patent/LT2591125T/lt unknown
- 2011-07-08 NO NO11736190A patent/NO2591125T3/no unknown
- 2011-07-08 EP EP18163123.5A patent/EP3360975B1/en active Active
- 2011-07-08 AU AU2011274642A patent/AU2011274642B2/en active Active
- 2011-07-08 DK DK11736190.7T patent/DK2591125T3/en active
- 2011-07-08 CN CN201180034117.6A patent/CN103180459B/zh active Active
- 2011-07-08 EP EP22164776.1A patent/EP4063518A1/en not_active Withdrawn
- 2011-07-08 ES ES11736190.7T patent/ES2667346T3/es active Active
- 2011-07-08 ES ES18163123T patent/ES2916339T3/es active Active
- 2011-07-08 KR KR1020137003437A patent/KR101862756B1/ko active Active
- 2011-07-08 NZ NZ606228A patent/NZ606228A/en unknown
- 2011-07-08 PL PL18163123T patent/PL3360975T3/pl unknown
- 2011-07-08 HR HRP20180593TT patent/HRP20180593T1/hr unknown
- 2011-07-08 RS RS20180488A patent/RS57295B1/sr unknown
- 2011-07-08 RU RU2013105459/10A patent/RU2603082C2/ru active
- 2011-07-08 PT PT117361907T patent/PT2591125T/pt unknown
- 2011-07-08 SM SM20180213T patent/SMT201800213T1/it unknown
-
2013
- 2013-01-07 IL IL224127A patent/IL224127B/en active IP Right Grant
- 2013-01-15 ZA ZA2013/00375A patent/ZA201300375B/en unknown
-
2017
- 2017-12-24 IL IL256509A patent/IL256509B/en active IP Right Grant
-
2018
- 2018-05-03 CY CY20181100457T patent/CY1121702T1/el unknown
-
2024
- 2024-06-10 US US18/738,818 patent/US20250066846A1/en active Pending
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| BR112013000552B1 (pt) | estratégias de sequenciamento de região de interesse genômica em 3d | |
| US20210363597A1 (en) | Identification and use of circulating nucleic acids | |
| JP2022001050A (ja) | ヌクレアーゼ、リガーゼ、ポリメラーゼ、及び配列決定反応の組み合わせを用いた、核酸配列、発現、コピー、またはdnaのメチル化変化の識別及び計数方法 | |
| US20180251845A1 (en) | Array-based translocation and rearrangement assays | |
| US20200131567A1 (en) | Methods of attaching adapters to sample nucleic acids | |
| US12371744B2 (en) | Method of sequencing a nucleic acid of interest | |
| WO2023018944A1 (en) | Methods for simultaneous mutation detection and methylation analysis | |
| HK1259754A1 (en) | 3-d genomic region of interest sequencing strategies | |
| HK1259754B (en) | 3-d genomic region of interest sequencing strategies | |
| US20180282799A1 (en) | Targeted locus amplification using cloning strategies | |
| Plongthongkum | Probing Interaction of Genome and Methylome by Targeted Bisulfite Sequencing |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| B08F | Application dismissed because of non-payment of annual fees [chapter 8.6 patent gazette] |
Free format text: REFERENTE A 4A ANUIDADE. |
|
| B08G | Application fees: restoration [chapter 8.7 patent gazette] | ||
| B06F | Objections, documents and/or translations needed after an examination request according [chapter 6.6 patent gazette] | ||
| B06T | Formal requirements before examination [chapter 6.20 patent gazette] | ||
| B07A | Application suspended after technical examination (opinion) [chapter 7.1 patent gazette] | ||
| B09A | Decision: intention to grant [chapter 9.1 patent gazette] | ||
| B16A | Patent or certificate of addition of invention granted [chapter 16.1 patent gazette] |
Free format text: PRAZO DE VALIDADE: 20 (VINTE) ANOS CONTADOS A PARTIR DE 08/07/2011, OBSERVADAS AS CONDICOES LEGAIS. |
|
| B25A | Requested transfer of rights approved |
Owner name: CERGENTIS B.V. (NL) |
|
| B25G | Requested change of headquarter approved |
Owner name: CERGENTIS B.V. (NL) |