KR20150042856A - 클라빈-유형 알칼로이드의 생산을 위한 유전자 및 방법 - Google Patents
클라빈-유형 알칼로이드의 생산을 위한 유전자 및 방법 Download PDFInfo
- Publication number
- KR20150042856A KR20150042856A KR1020157006881A KR20157006881A KR20150042856A KR 20150042856 A KR20150042856 A KR 20150042856A KR 1020157006881 A KR1020157006881 A KR 1020157006881A KR 20157006881 A KR20157006881 A KR 20157006881A KR 20150042856 A KR20150042856 A KR 20150042856A
- Authority
- KR
- South Korea
- Prior art keywords
- seq
- polypeptide
- amino acid
- polynucleotide
- sequence identity
- Prior art date
Links
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/0004—Oxidoreductases (1.)
- C12N9/001—Oxidoreductases (1.) acting on the CH-CH group of donors (1.3)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12R—INDEXING SCHEME ASSOCIATED WITH SUBCLASSES C12C - C12Q, RELATING TO MICROORGANISMS
- C12R2001/00—Microorganisms ; Processes using microorganisms
- C12R2001/01—Bacteria or Actinomycetales ; using bacteria or Actinomycetales
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N1/00—Microorganisms, e.g. protozoa; Compositions thereof; Processes of propagating, maintaining or preserving microorganisms or compositions thereof; Processes of preparing or isolating a composition containing a microorganism; Culture media therefor
- C12N1/20—Bacteria; Culture media therefor
- C12N1/205—Bacterial isolates
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P17/00—Preparation of heterocyclic carbon compounds with only O, N, S, Se or Te as ring hetero atoms
- C12P17/10—Nitrogen as only ring hetero atom
- C12P17/12—Nitrogen as only ring hetero atom containing a six-membered hetero ring
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K31/00—Medicinal preparations containing organic active ingredients
- A61K31/33—Heterocyclic compounds
- A61K31/395—Heterocyclic compounds having nitrogen as a ring hetero atom, e.g. guanethidine or rifamycins
- A61K31/435—Heterocyclic compounds having nitrogen as a ring hetero atom, e.g. guanethidine or rifamycins having six-membered rings with one nitrogen as the only ring hetero atom
- A61K31/47—Quinolines; Isoquinolines
- A61K31/48—Ergoline derivatives, e.g. lysergic acid, ergotamine
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07D—HETEROCYCLIC COMPOUNDS
- C07D209/00—Heterocyclic compounds containing five-membered rings, condensed with other rings, with one nitrogen atom as the only ring hetero atom
- C07D209/56—Ring systems containing three or more rings
- C07D209/80—[b, c]- or [b, d]-condensed
- C07D209/90—Benzo [c, d] indoles; Hydrogenated benzo [c, d] indoles
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07D—HETEROCYCLIC COMPOUNDS
- C07D487/00—Heterocyclic compounds containing nitrogen atoms as the only ring hetero atoms in the condensed system, not provided for by groups C07D451/00 - C07D477/00
- C07D487/02—Heterocyclic compounds containing nitrogen atoms as the only ring hetero atoms in the condensed system, not provided for by groups C07D451/00 - C07D477/00 in which the condensed system contains two hetero rings
- C07D487/06—Peri-condensed systems
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/195—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from bacteria
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/37—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from fungi
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/14—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from fungi, algea or lichens
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/40—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against enzymes
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N1/00—Microorganisms, e.g. protozoa; Compositions thereof; Processes of propagating, maintaining or preserving microorganisms or compositions thereof; Processes of preparing or isolating a composition containing a microorganism; Culture media therefor
- C12N1/14—Fungi; Culture media therefor
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N1/00—Microorganisms, e.g. protozoa; Compositions thereof; Processes of propagating, maintaining or preserving microorganisms or compositions thereof; Processes of preparing or isolating a composition containing a microorganism; Culture media therefor
- C12N1/14—Fungi; Culture media therefor
- C12N1/145—Fungal isolates
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N1/00—Microorganisms, e.g. protozoa; Compositions thereof; Processes of propagating, maintaining or preserving microorganisms or compositions thereof; Processes of preparing or isolating a composition containing a microorganism; Culture media therefor
- C12N1/14—Fungi; Culture media therefor
- C12N1/16—Yeasts; Culture media therefor
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N1/00—Microorganisms, e.g. protozoa; Compositions thereof; Processes of propagating, maintaining or preserving microorganisms or compositions thereof; Processes of preparing or isolating a composition containing a microorganism; Culture media therefor
- C12N1/20—Bacteria; Culture media therefor
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/52—Genes encoding for enzymes or proenzymes
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/80—Vectors or expression systems specially adapted for eukaryotic hosts for fungi
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/0004—Oxidoreductases (1.)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/10—Transferases (2.)
- C12N9/1003—Transferases (2.) transferring one-carbon groups (2.1)
- C12N9/1007—Methyltransferases (general) (2.1.1.)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/10—Transferases (2.)
- C12N9/1085—Transferases (2.) transferring alkyl or aryl groups other than methyl groups (2.5)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/90—Isomerases (5.)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P1/00—Preparation of compounds or compositions, not provided for in groups C12P3/00 - C12P39/00, by using microorganisms or enzymes
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P17/00—Preparation of heterocyclic carbon compounds with only O, N, S, Se or Te as ring hetero atoms
- C12P17/10—Nitrogen as only ring hetero atom
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P17/00—Preparation of heterocyclic carbon compounds with only O, N, S, Se or Te as ring hetero atoms
- C12P17/18—Preparation of heterocyclic carbon compounds with only O, N, S, Se or Te as ring hetero atoms containing at least two hetero rings condensed among themselves or condensed with a common carbocyclic ring system, e.g. rifamycin
- C12P17/182—Heterocyclic compounds containing nitrogen atoms as the only ring heteroatoms in the condensed system
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N2523/00—Culture process characterised by temperature
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12R—INDEXING SCHEME ASSOCIATED WITH SUBCLASSES C12C - C12Q, RELATING TO MICROORGANISMS
- C12R2001/00—Microorganisms ; Processes using microorganisms
- C12R2001/645—Fungi ; Processes using fungi
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12Y—ENZYMES
- C12Y205/00—Transferases transferring alkyl or aryl groups, other than methyl groups (2.5)
- C12Y205/01—Transferases transferring alkyl or aryl groups, other than methyl groups (2.5) transferring alkyl or aryl groups, other than methyl groups (2.5.1)
-
- Y—GENERAL TAGGING OF NEW TECHNOLOGICAL DEVELOPMENTS; GENERAL TAGGING OF CROSS-SECTIONAL TECHNOLOGIES SPANNING OVER SEVERAL SECTIONS OF THE IPC; TECHNICAL SUBJECTS COVERED BY FORMER USPC CROSS-REFERENCE ART COLLECTIONS [XRACs] AND DIGESTS
- Y02—TECHNOLOGIES OR APPLICATIONS FOR MITIGATION OR ADAPTATION AGAINST CLIMATE CHANGE
- Y02P—CLIMATE CHANGE MITIGATION TECHNOLOGIES IN THE PRODUCTION OR PROCESSING OF GOODS
- Y02P20/00—Technologies relating to chemical industry
- Y02P20/50—Improvements relating to the production of bulk chemicals
- Y02P20/52—Improvements relating to the production of bulk chemicals using catalysts, e.g. selective catalysts
Landscapes
- Chemical & Material Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Health & Medical Sciences (AREA)
- Organic Chemistry (AREA)
- Engineering & Computer Science (AREA)
- Genetics & Genomics (AREA)
- Wood Science & Technology (AREA)
- Zoology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Biotechnology (AREA)
- General Health & Medical Sciences (AREA)
- Biochemistry (AREA)
- General Engineering & Computer Science (AREA)
- Microbiology (AREA)
- Biomedical Technology (AREA)
- Molecular Biology (AREA)
- Medicinal Chemistry (AREA)
- Biophysics (AREA)
- Chemical Kinetics & Catalysis (AREA)
- General Chemical & Material Sciences (AREA)
- Mycology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Physics & Mathematics (AREA)
- Plant Pathology (AREA)
- Virology (AREA)
- Tropical Medicine & Parasitology (AREA)
- Immunology (AREA)
- Botany (AREA)
- Gastroenterology & Hepatology (AREA)
- Pharmacology & Pharmacy (AREA)
- Epidemiology (AREA)
- Animal Behavior & Ethology (AREA)
- Public Health (AREA)
- Veterinary Medicine (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Preparation Of Compounds By Using Micro-Organisms (AREA)
- Enzymes And Modification Thereof (AREA)
- Peptides Or Proteins (AREA)
Abstract
본 발명은 원칙적으로 클라빈-유형 알칼로이드, 예컨대 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 및 카노클라빈 알데히드의 재조합 제조의 분야에 속한다. 본 발명은 클라빈-유형 알칼로이드의 제조를 위한 방법에 적용될 수 있는 클라빈-유형 알칼로이드 및 폴리펩티드, 폴리뉴클레오티드 및 상기 폴리뉴클레오티드를 포함하는 벡터의 제조를 위한 미생물 및 공정을 제공한다.
Description
본원은 그 전문을 본원에 참조로 포함하는 US 61/691848, EP 12181388.5, US 61/786841, EP 13159444.2 및 EP 13178008.2를 기초로 한 우선권을 주장한다.
발명의 분야
본 발명은 원칙적으로 클라빈-유형 알칼로이드, 예컨대 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 및 카노클라빈 알데히드의 재조합 제조의 분야에 속한다. 본 발명은 클라빈-유형 알칼로이드의 제조를 위한 방법에 적용될 수 있는 클라빈-유형 알칼로이드 및 폴리펩티드, 폴리뉴클레오티드 및 상기 폴리뉴클레오티드를 포함하는 벡터의 제조를 위한 미생물 및 공정을 제공한다.
맥각 알칼로이드는 다양한 구조 및 생리학적 활성을 갖는 복잡한 패밀리의 인돌 유도체이고 ([Flieger 1997, Folia Microbiol (Praha) 42:3-30]; [Schardl 2006, Chem Biol 63:45-86]), 클라비시피타세아에(Clavicipitaceae) (예를 들어 클라비셉스(Claviceps) 및 네오티포디움(Neotyphodium) 또는 에피클로에(Epichloee)) 및 트리코코마세아에(Trichocomaceae) (아스페르길루스(Aspergillus) 및 페니실리움(Penicillium) 포함) 과의 진균에 의해 생산된다. 클라비셉스, 페니실리움, 및 아스페르길루스 속의 진균이 중요한 천연 생산자이지만 ([Flieger 1997, 상기 문헌]; [Schradl 상기 문헌]), 스파셀리아(Sphacelia), 발란시아(Balansia) 또는 페리글란둘라(Periglandula) 속에서도 또한 발견된다 ([Pazoutova, S. et al., 2008, Fungal Diversity, 31: 95-110] 및 [Steiner, U. 2011, Mycologia, 103(5):1133-1145]). 천연 맥각 알칼로이드 및 그들의 반합성 유도체는 모두 현대 의학에서 널리 사용되고, 자궁수축 활성, 혈압의 조정, 뇌하수체 호르몬의 분비의 제어, 편두통 예방, 및 도파민작용성 및 신경이완 활성을 비롯한 광범위한 약물학적 활성을 나타낸다 ([de Groot 1998, Drugs 56:523-535]; [Haarmann 2009, Mol Plant Pathol 10:563-577]; [Schardl 2006, 상기 문헌]). 맥각 알칼로이드는 그들의 구조적 특징에 따라 2개의 클래스로 나누어질 수 있으며, 즉 D-리세르그산의 아미드 유도체 및 클라빈-유형 알칼로이드 ([Flieger 1997, 상기 문헌];, [Groger 1998, Alkaloids Chem Biol 50:171-218]). 첫 번째 군의 구성원은 대체로 리세르그산 및 펩티드 모이어티로 구성된다. 이들은 또한 에르고펩틴으로도 칭해진다. 에르고펩틴의 중요한 구성원은 예를 들어, 에르고타민 및 에르고발린이다. 클라빈-유형 알칼로이드, 예를 들어 시클로클라빈, 아그로클라빈, 푸미가클라빈 및 유사한 물질, 예를 들어 엘리모클라빈, 피로클라빈, 코스타클라빈 또는 엡코스타클라빈 및 그들의 전구체 카노클라빈-I 및 카노클라빈 알데히드는 단지 에르고펩틴처럼 동일한 또는 유사한 트리시클릭 또는 테트라시클릭 고리 시스템으로만 이루어지지만, 펩티드 모이어티는 결핍된다. 그러나, 클라빈-유형 알칼로이드는 또한 추가의 치환기를 포함할 수 있고, 따라서 상기 언급된 클라빈-유형 알칼로이드에 제한되지는 않는다. 상기 유도체의 예는 푸미가클라빈의 군이고, 예를 들어 푸미클라빈 A, B 또는 C, 이소푸미가클라빈 A 또는 B, 또는 9-데아세틸푸미가클라인 C (Wallwey, C. and Li, S.M. 2011, Nat. Prod. Rep., 28:496-510)는 페스투클라빈과 기본 구조를 공유하지만, 추가의 치환기를 포함한다. 푸미가클라빈은 예를 들어, 페니실리움 및 아스페르길루스, 예를 들어, 에이. 푸미가투스(A. fumigatus)에 의해 생산된다 (Flieger 1997, 상기 문헌). 그러나, 클라비시피타세아에의 진균 과, 예를 들어 씨. 푸르푸레아(C. purpurea) (Flieger 1997, 상기 문헌)는 리세르그산의 유도체인 것으로 간주될 수 있는 에르고펩틴을 생산하는 능력을 갖고, 그의 전구체 중 하나는 클라빈-유형 알칼로이드 아그로클라빈이다. 에르고펩티드 및 클라빈-유형 알칼로이드의 전구체를 비교하면 그들의 생합성 경로의 초기 단계는 매우 유사하고, 마찬가지로 예를 들어 에이. 푸미가투스 및 씨. 푸르푸레아에 의해 공유되는 반면, 경로의 나중의 단계는 2개의 진균 종에서 상이함을 가르킨다 ([Li 2006, Chembiochem 7:158-164]; [Panaccione 2005, FEMS Microbiol Lett 251:9-17]; [Schardl 2006, 상기 문헌]).
클라빈-유형 알칼로이드 및 심지어 맥각 알칼로이드의 발효 생산은 일반적으로 맥각 알칼로이드의 천연 생산자가 대규모 배양하기 어려운 경향이 있다는 문제를 극복해야 한다. 이에 대한 하나의 이유는 사상 진균을 침수 배양으로 발효시키는 기술적 문제이다. 일부 천연 맥각 알칼로이드 생산자는 심지어 식물 속, 예를 들어 이포모에아(Ipomoea), 투르비나(Turbina), 아르기레이아(Argyreia) 및 스트릭토카르디아(Strictocardia) (예를 들어, 페리글란둘라 종) 또는 특정 초본 종, 예를 들어 롤리움(Lolium), 소르굼(Sorghum) 또는 페스투카(Festuca) (예를 들어 네오티포디움 및 에피클로에 종)의 내부 기생식물이다. 또한, 많은 천연 맥각 알칼로이드 생산자는 이들 알칼로이드를 단지 특수한 조건 하에서 또는 특정 발생기 동안에 생산하고, 심지어 대체로 낮은 생산 속도로 생산한다. 많은 천연 맥각 알칼로이드 생산자는 상이한 맥각 알칼로이드들의 집합체를 생산하고, 이것은 주어진 알칼로이드를 비용 효과적인 방식으로 단리하는데 문제를 일으키고, 관심있는 특정 알칼로이드의 생산을 더욱 저하시킨다. 맥각 알칼로이드 또는 훨씬 덜 복잡한 클라빈-유형 알칼로이드의 총 화학적 합성은 지금까지, 여전히 상당한 도전 과제를 갖고, 대체로 라세미 혼합물의 합성을 일으킨다.
이들 문제를 극복하기 위해, 본 발명은 재조합 미생물 및 재조합 천연 맥각 알칼로이드 생산자 유기체, 및 폴리뉴클레오티드, 폴리펩티드, 벡터, 및 이들 재조합 미생물 및 재조합 천연 맥각 알칼로이드 생산자 유기체를 사용하는 방법, 및 클라빈-유형 알칼로이드의 생산을 위한 폴리뉴클레오티드, 폴리펩티드 및 벡터를 제공한다.
본 발명은
a) 적어도 하나의 EasH 활성, 또는
b) 적어도 하나의 EasH 및 EasD 활성, 또는
c) 적어도 하나의 EasH 및 EasA 리덕타제 활성, 또는
d) 적어도 하나의 EasH, EasD 및 EasA 활성, 또는
e) 적어도 하나의 EasH 및 EasD, EasA 리덕타제 및 EasG 활성, 또는
f) 적어도 하나의 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 및 EasG 활성, 또는
g) 적어도 하나의 EasD, EasA 및 EasG 활성, 또는
h) 적어도 하나의 DmaW, EasF, EasE, EasC, EasD, EasA 리덕타제 및 EasG 활성, 또는
i) 적어도 하나의 DmaW, EasF, EasE, EasC, EasD, EasA 이소머라제 및 EasG 활성, 또는
j) 적어도 하나의 DmaW, EasF, EasE 및 EasC 활성,
k) a) 내지 j) 중 적어도 2개의 조합
을 포함하는 재조합 미생물을 제공하고,
여기서 재조합 미생물은 천연 맥각 알칼로이드 생산자 유기체가 아니다.
본 발명은 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 및 EasG 활성으로 이루어지는 활성의 군으로부터 선택되는 적어도 하나의 상향조절된 활성을 갖는 추가의 재조합 천연 맥각 알칼로이드 생산자 유기체를 제공하고, 여기서 EasA 활성은 EasA 리덕타제, EasA 이소머라제 또는 EasA 리덕타제 및 EasA 이소머라제 활성이다.
재조합 천연 맥각 알칼로이드 생산자 유기체는
a) 적어도 하나의 EasH 활성, 또는
b) 적어도 하나의 EasH 및 EasD, 또는
c) 적어도 하나의 EasH 및 EasA 리덕타제 활성, 또는
d) 적어도 하나의 EasH, EasD 및 EasA 활성, 또는
e) 적어도 하나의 EasH 및 EasD, EasA 리덕타제 및 EasG 활성, 또는
f) 적어도 하나의 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 및 EasG 활성, 또는
g) 적어도 하나의 EasD, EasA 및 EasG 활성, 또는
h) 적어도 하나의 DmaW, EasF, EasE, EasC, EasD, EasA 리덕타제 및 EasG 활성, 또는
i) 적어도 하나의 DmaW, EasF, EasE, EasC, EasD, EasA 이소머라제 및 EasG 활성, 또는
j) 적어도 하나의 DmaW, EasF, EasE 및 EasC 활성
의 군 중의 하나 이상의 활성으로부터 선택되는 적어도 하나의 상향조절된 활성을 포함한다.
추가로, EasD, EasH, EasA 리덕타제, EasA 이소머라제 및 EasG 활성의 군으로부터 선택되는 적어도 하나의 하향조절된 활성을 갖는 상기 설명한 재조합 천연 맥각 알칼로이드 생산자 유기체가 제공된다. 본 발명의 또 다른 실시양태는 DMAPP의 확대된 공급 및/또는 Me-DMAT의 확대된 공급을 포함하는, 바람직하게는 적어도 하나의 하향조절된 ERG9 또는 ERG20 활성, 또는 적어도 하나의 상향조절된 HMG-CoA 리덕타제 활성을 포함하는, 또는 적어도 하나의 하향조절된 ERG9 또는 ERG20 활성 및 적어도 하나의 상향조절된 HMG-CoA 리덕타제 활성을 포함하는, 본원에서 설명되는 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체이다.
본 발명의 다른 실시양태는 다음을 코딩하는 적어도 하나의 재조합 폴리뉴클레오티드를 포함하는, 상기 설명한 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체를 포함한다:
a) 적어도 하나의 EasH 활성, 또는
b) 적어도 하나의 EasH 및 EasD 활성, 또는
c) 적어도 하나의 EasA 리덕타제 또는 적어도 하나의 EasA 이소머라제 활성, 또는
d) 적어도 하나의 EasD, EasA 이소머라제 및 EasG 활성, 또는
e) 적어도 하나의 EasD, EasA 리덕타제 및 EasG 활성, 또는
f) 적어도 하나의 EasH, EasD, EasA 리덕타제, EasA 이소머라제 및 EasG 활성, 또는
g) 적어도 하나의 EasH, EasD, EasA 리덕타제 및 EasG 활성, 또는
h) 적어도 하나의 EasH, EasD, EasA 이소머라제 및 EasG 활성, 또는
i) 적어도 하나의 DmaW, EasF, EasE 및 EasC 활성, 또는
j) a) 내지 i) 중 적어도 2개의 조합.
본 발명은 또한 다음을 코딩하는 재조합 폴리뉴클레오티드를 제공한다:
a) 적어도 하나의 EasH 활성, 또는
b) 적어도 하나의 EasH 및 EasD 활성, 또는
c) 적어도 하나의 EasA 리덕타제 또는 적어도 하나의 EasA 이소머라제 활성, 또는
d) 적어도 하나의 EasD, EasA 이소머라제 및 EasG 활성, 또는
e) 적어도 하나의 EasD, EasA 리덕타제 및 EasG 활성, 또는
f) 적어도 하나의 EasH, EasD, EasA 리덕타제, EasA 이소머라제 및 EasG 활성, 또는
g) 적어도 하나의 EasH, EasD, EasA 리덕타제 및 EasG 활성, 또는
h) 적어도 하나의 EasH, EasD, EasA 이소머라제 및 EasG 활성, 또는
i) 적어도 하나의 DmaW, EasF, EasE 및 EasC 활성
j) a) 내지 i) 중 적어도 2개의 조합.
추가로, DmaW 활성이 다음에 의한 것인, 상기 설명한 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체, 또는 설명된 재조합 폴리뉴클레오티드를 제공한다:
a) 서열 145 및 146에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
b) 서열 147 또는 190에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
c) 서열 1, 19, 20, 179, 또는 180에 적어도 40% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 1, 19, 20, 21, 22, 23, 24, 25, 26, 27 또는 28에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
e) 서열 102, 122 또는 123에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
f) 서열 129 또는 137에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는,
g) 서열 102, 122 또는 123에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
h) 엄격한 조건 하에 서열 102, 122 또는 123에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
i) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 h) 중 적어도 2개, 바람직하게는 적어도 b) 및 c)에 따른 폴리펩티드, 또는 폴리펩티드 a) 내지 i) 중 적어도 하나에 따른 폴리펩티드.
추가의 실시양태는 EasF 활성이 다음에 의한 것인, 본원에서 설명되는 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체, 또는 상기 설명한 재조합 폴리뉴클레오티드를 포함한다:
a) 서열 154에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
b) 서열 6, 75 또는 76에 적어도 40% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
c) 서열 6, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84 또는 85에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 107 또는 120에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
e) 서열 134 또는 142에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는,
f) 서열 107 또는 120에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
g) 엄격한 조건 하에 서열 107 또는 120에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
h) a) 내지 g) 중 적어도 2개, 바람직하게는 적어도 a) 및 b)에 따른 폴리펩티드, 또는
i) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 h) 중 적어도 하나에 따른 폴리펩티드.
본 발명의 다른 부분은 EasE 활성이 다음에 의한 것인, 상기 설명한 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체, 또는 상기 설명한 재조합 폴리뉴클레오티드를 포함한다:
a) 서열 153, 185 또는 191에 적어도 90% 서열 동일성, 바람직하게는 서열 191에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는,
b) 서열 5 또는 64에 적어도 40% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
c) 서열 5, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73 또는 74에 적어도 90% 서열 동일성을 갖는, 또는 서열 5, 64, 175, 66, 67, 68, 69, 176, 71, 177, 73 또는 178에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 106에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
e) 서열 133 또는 141에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는,
f) 서열 106, 133 또는 141에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드 또는
g) 엄격한 조건 하에 서열 106, 133 또는 141에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드 또는
h) a) 내지 g) 중 적어도 2개, 바람직하게는 적어도 a) 및 b)에 따른 폴리펩티드, 또는
i) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 h) 중 적어도 하나에 따른 폴리펩티드.
추가의 부분은 EasC 활성이 다음에 의한 것인, 상기 설명한 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체, 또는 상기 설명한 재조합 폴리뉴클레오티드를 포함한다:
a) 서열 151 또는 192에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는,
b) 서열 3 또는 41에 적어도 40% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
c) 서열 3, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51 또는 52에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 104에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
e) 서열 131 또는 139에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는,
f) 서열 104, 131 또는 139에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
g) 엄격한 조건 하에 서열 104, 131 또는 139에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
h) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 g) 중 적어도 2개, 바람직하게는 적어도 a) 및 b)에 따른 폴리펩티드, 또는 a) 내지 h) 중 적어도 하나에 따른 폴리펩티드.
본 발명은 EasD 활성이 다음에 의한 것인, 상기 설명한 추가의 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체, 또는 상기 설명한 재조합 폴리뉴클레오티드를 포함한다:
a) 서열 152에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는,
b) 서열 4 또는 53에 적어도 40% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
c) 서열 4, 53, 54, 55, 56, 57, 58, 59, 60, 62 또는 63에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 105에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
e) 서열 132 또는 140에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
f) 서열 105, 132 또는 140에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
g) 엄격한 조건 하에 서열 105, 132 또는 140에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
h) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 g) 중 적어도 2개, 바람직하게는 적어도 a) 및 b)에 따른 폴리펩티드, 또는 a) 내지 h) 중 적어도 하나에 따른 폴리펩티드.
본 발명의 다른 부분은 EasH 활성이 다음에 의한 것인, 상기 설명한 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체, 또는 상기 설명한 재조합 폴리뉴클레오티드를 포함한다:
a) 서열 156에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
b) 서열 8 및/또는 95에 적어도 60% 서열 동일성을 갖고 서열 156에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
c) 서열 8 및/또는 95에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 109, 및/또는 157에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
e) 서열 109, 136, 144 및/또는 157에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는,
f) 서열 109, 136, 144 및/또는 157에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드 또는
g) 엄격한 조건 하에 서열 109, 136, 144 및/또는 157에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드 또는
h) a) 내지 g) 중 적어도 2개에 따른 폴리펩티드, 또는
i) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 h) 중 적어도 하나에 따른 폴리펩티드.
추가의 실시양태는 EasA 리덕타제 활성이 다음에 의한 것인, 상기 설명한 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체, 또는 상기 설명한 재조합 폴리뉴클레오티드를 포함한다:
a) 서열 148, 149 및 150에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하고 서열 149의 아미노산 위치 18에 티로신을 갖는 폴리펩티드, 또는
b) 서열 2 또는 31에 적어도 40% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
c) 서열 2, 31, 32, 33, 34, 35, 36, 37, 38, 39 또는 40에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 103에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
e) 서열 130 또는 138에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는,
f) 서열 103에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
g) 엄격한 조건 하에 서열 103에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
h) a) 내지 g) 중 적어도 2개, 바람직하게는 적어도 a) 및 b)에 따른 폴리펩티드, 또는
i) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 h) 중 적어도 하나에 따른 폴리펩티드.
다른 실시양태는 EasA 이소머라제 활성이 다음에 의한 것인, 상기 설명한 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체, 또는 상기 설명한 재조합 폴리뉴클레오티드를 포함한다:
a) 서열 148, 149 및 150에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하고 서열 149의 아미노산 위치 18에 페닐알라닌을 갖는 폴리펩티드, 또는
b) 서열 2 또는 31에 적어도 40% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
c) 서열 2, 31, 32, 33, 34, 35, 36, 37, 38, 39 또는 40에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 103에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
e) 서열 130 또는 138에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는,
f) 서열 103에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
g) 엄격한 조건 하에 서열 103에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
h) a) 내지 g) 중 적어도 2개, 바람직하게는 적어도 a) 및 b)에 따른 폴리펩티드, 또는
i) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 h) 중 적어도 하나에 따른 폴리펩티드.
추가의 실시양태는 EasG 활성이 다음에 의한 것인, 상기 설명한 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체, 또는 상기 설명한 재조합 폴리뉴클레오티드를 포함한다:
a) 서열 155 또는 183에 적어도 90% 서열 동일성, 바람직하게는 서열 183에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는,
b) 서열 7 또는 86에 적어도 39% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
c) 서열 7, 86, 87, 88, 89, 90, 91, 92, 93 또는 94에 적어도 70% 서열 동일성, 바람직하게는 서열 7, 86, 87, 88, 89, 90, 181, 92, 93 또는 94에 적어도 70% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 108에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
e) 서열 135 또는 143에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
f) 서열 108에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
g) 엄격한 조건 하에 서열 108에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
h) a) 내지 g) 중 적어도 2개, 바람직하게는 적어도 a) 및 b)에 따른 폴리펩티드, 또는
i) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 h) 중 적어도 하나에 따른 폴리펩티드.
본 발명은 ERG9 활성이 서열 9에 적어도 70% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드에 의한 것이거나, 또는 ERG20 활성이 서열 10에 적어도 70% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드에 의한 것이거나, 또는 HMG-CoA 리덕타제 활성이 서열 11에 적어도 70% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드에 의한 것이거나, 또는 ERG9, ERG20 및 HMG-CoA 리덕타제 활성 중 적어도 2개가 서열 9, 10 또는 11에 적어도 70% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드에 의한 것이거나, 또는 ERG9, ERG20 및 HMG-CoA 리덕타제 활성이 서열 9, 10 또는 11에 적어도 70% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드에 의한 것인, 상기 설명한 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체를 추가로 제공한다. 추가의 실시양태는
a) 시클로클라빈,
b) 페스투클라빈,
c) 아그로클라빈,
d) 카노클라빈 알데히드,
e) 카노클라빈 I
의 화합물의 군으로부터 선택되는 적어도 하나의 화합물을 포함하는 재조합 미생물을 포함하고, 여기서 재조합 미생물은 천연 맥각 알칼로이드 생산자 유기체가 아니다.
본 발명은 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및 카노클라빈 I의 군으로부터 선택되는 적어도 하나의 화합물을 동일한 조건 하에 성장할 때 비-재조합 야생형 유기체에 비해 보다 많은 양으로 포함하고/하거나 생산하는 재조합 천연 맥각 알칼로이드 생산자 유기체를 제공한다. 추가의 실시양태는
a) 시클로클라빈,
b) 페스투클라빈,
c) 아그로클라빈,
d) 카노클라빈 알데히드,
e) 카노클라빈 I
의 화합물의 군으로부터 선택되는 적어도 하나의 화합물을 포함하는, 상기 설명한 본 발명의 부분 및 실시양태의 임의의 하나에서 설명되는 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체를 포함한다.
본 발명의 다른 부분은 본 발명의 부분 및 실시양태의 임의의 하나에서 설명되는 폴리펩티드를 코딩하는 적어도 하나의 폴리뉴클레오티드의 발현을 위한 재조합 발현 카세트 또는 적어도 하나의 상기 발현 카세트를 포함하는 폴리뉴클레오티드를 포함한다. 본 발명의 추가의 부분은
a) 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체에서 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나의 생산을 허용하는 조건 하에 상기 설명한 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체를 배양하는 단계; 및
b) 상기 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및/또는 카노클라빈 I을 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 및/또는 배양 배지로부터 얻는 단계
를 포함하는, 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나의 생산 방법이다.
본 발명의 다른 방법은
a) 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체에서 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나의 생산을 허용하는 조건 하에 상기 설명한 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체를 배양하는 단계;
b) 상기 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체에게 IPP, 트립토판, DMAPP, DMAT, Me-DMAT 또는 카노클라빈 I의 군으로부터 선택되는 적어도 하나의 화합물을 배양 배지를 통해 제공하는 단계; 및
c) 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 및/또는 배양 배지로부터 상기 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I을 얻는 단계
를 포함하는, 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나의 생산 방법을 제공한다.
상기 설명한 방법의 변형은 시클로클라빈이 생산되고 얻어지는, 또는 페스투클라빈이 생산되고 얻어지는 또는 아그로클라빈이 생산되고 얻어지는, 또는 카노클라빈 I이 배지에 공급되고 시클로클라빈이 생산되고 얻어지는, 또는 카노클라빈 I이 생산되고 얻어지는, 또는 카노클라빈 알데히드가 생산되고 얻어지는 방법을 포함한다. 상기 설명한 방법은 상이한 배양 온도를 이용할 수 있다. 따라서, 본 발명의 추가의 부분은 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체가 10℃ 내지 40℃, 바람직하게는 15℃ 내지 35℃, 보다 바람직하게는 18℃ 내지 32℃, 보다 더 바람직하게는 20℃ 내지 30℃의 온도에서 배양되는 방법을 포함한다. 방법은 10℃ 내지 32℃, 13℃ 내지 32℃, 15℃ 내지 32℃, 16℃ 내지 32℃, 17℃ 내지 32℃, 18℃ 내지 32℃, 19℃ 내지 32℃, 20℃ 내지 32℃, 15℃ 내지 31℃, 15℃ 내지 30℃, 15℃ 내지 29℃, 15℃ 내지 28℃, 15℃ 내지 27℃, 15℃ 내지 26℃, 15℃ 내지 25℃의 온도에서 배양된 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체를 이용할 수 있다. 상기 방법의 변형은 배양 온도의 변화를 포함한다. 따라서, 본 발명은 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체가 먼저 25℃ 내지 40℃, 바람직하게는 25℃ 내지 35℃의 온도에서, 이어서 10℃ 내지 25℃, 바람직하게는 15℃ 내지 25℃의 온도에서 배양되는, 또는 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체가 먼저 10℃ 내지 25℃, 바람직하게는 15℃ 내지 25℃의 온도에서, 이어서 25℃ 내지 40℃, 바람직하게는 25℃ 내지 35℃의 온도에서 배양되는 상기 설명한 방법을 제공한다. 또한, 본 발명은 적어도 하나의 생산된 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I이 추출을 통해 얻어지는 상기 설명한 방법을 제공한다. 상기 방법은 적어도 하나의 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I이 에틸 아세테이트 (EA), tert-부틸 메틸에테르 (TBME), 클로로포름 (CHCl3) 및 디클로로메탄 (DCM)의 군으로부터 선택되는 화합물을 사용한 추출을 포함하는 추출 방법을 통해 얻어지는 방법 및 적어도 하나의 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I이 pH=10에서 에틸 아세테이트를 사용한 액체/액체 추출, 바람직하게는 pH가 NaOH로 조정되는, pH=10에서 에틸 아세테이트를 사용한 액체/액체 추출을 포함하는 추출 방법을 통해 얻어지는 방법을 포함한다. 상기 방법의 추가의 변형은 적어도 하나의 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I이 바람직하게는 pH=3의 HLB 수지, pH=3의 디아이온(Diaion) 수지, pH=3의 앰벌라이트(Amberlite) 1180 수지, pH=10의 앰벌라이트 1180 수지, pH=3의 앰벌라이트 16N 수지 또는 pH=10의 앰벌라이트 16N 수지의 군으로부터 선택되는 실리카계 수지 물질을 사용한 고상 추출을 사용한 추출을 포함하는 추출 방법을 통해 얻어지는 방법을 포함한다. 본 발명의 추가의 부분은 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I의 군으로부터 선택되는 적어도 하나의 화합물의 생산을 위한 또는 상기 설명한 임의의 방법에서, 상기 설명한 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체의 용도 또는 상기 설명한 재조합 폴리뉴클레오티드의 용도, 또는 상기 설명한 벡터의 용도를 포함한다. 본 발명은 상기 설명한 임의의 방법에 의해 생산된 추가의 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I을 포함한다. 본 발명의 다른 부분은 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나를 포함하지만, 다른 맥각 알칼로이드는 존재하지 않거나 낮은 함량으로 존재하는, 상기 설명한 방법에서 생산된 배양 배지, 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나를 포함하지만, 다른 맥각 알칼로이드는 존재하지 않거나 낮은 함량으로 존재하는 배양 배지이다. 본 발명은 상기 설명한 본 발명의 임의의 일부 및 실시양태에서 사용될 수 있는 추가의 폴리뉴클레오티드를 추가로 제공한다.
따라서, 본 발명은 다음으로 이루어지는 군으로부터 선택되는 하나 이상의 핵산 서열을 포함하는 폴리뉴클레오티드를 포함한다:
a) 서열 129, 또는 130, 131, 132, 133, 134, 135 또는 136에 제시된 뉴클레오티드 서열을 갖는 핵산 서열;
b) 서열 20, 31, 41, 53, 64, 75, 86 또는 95에 제시된 아미노산 서열을 갖는 폴리펩티드를 코딩하는 핵산 서열;
c) 서열 129, 또는 130, 131, 132, 133, 134, 135 또는 136에 제시된 핵산 서열에 적어도 70% 동일하고 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 리덕타제, EasA 이소머라제 또는 EasG 활성을 갖는 폴리펩티드를 코딩하는 핵산 서열;
d) DmaW, EasF, EasE, EasC, EasD, EasH, EasA 리덕타제, EasA 이소머라제 또는 EasG 활성을 갖고 서열 20, 31, 41, 53, 64, 75, 86 또는 95에 제시된 아미노산 서열에 적어도 70% 동일한 아미노산 서열을 갖는 폴리펩티드를 코딩하는 핵산 서열; 및
e) 엄격한 조건 하에 a) 내지 d) 중 임의의 하나에 혼성화할 수 있고 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 리덕타제, EasA 이소머라제 또는 EasG 활성을 갖는 폴리펩티드를 코딩하는 핵산 서열.
본 발명의 추가의 부분은 폴리뉴클레오티드가 상기 설명한 본 발명의 임의의 부분 또는 실시양태에서 설명된 폴리펩티드를 코딩하는 적어도 하나의 핵산 서열을 추가로 포함하는 상기 설명한 폴리뉴클레오티드를 추가로 포함한다. 본 발명의 추가의 실시양태는 상기 폴리뉴클레오티드를 포함하는 벡터 또는 발현 카세트 및 이들 발현 카세트 및/또는 본 발명에서 설명된 임의의 하나의 폴리펩티드에 대한 발현 카세트를 포함하는 벡터를 포함한다. 본 발명의에 포함되는 임의의 하나의 폴리펩티드의 발현을 위한 발현 벡터 및 상기 벡터 또는 상기 설명한 적어도 하나의 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 숙주 세포가 본 발명에 추가로 포함된다. 이들 숙주 세포는 박테리아 세포, 진균 세포, 특히 사상 진균 세포, 또는 효모 세포일 수 있다. 본 발명은
a) 상기 설명한 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드의 생산을 허용하는 조건 하에 상기 설명한 숙주 세포 또는 파에실로미세스 디바리카투스(Paecilomyces divaricatus) 세포를 배양하는 단계; 및
b) 단계 a)의 숙주 세포로부터 폴리펩티드를 얻는 단계
를 포함하는, 상기 설명한 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드의 제조를 위한 방법을 추가로 제공한다.
따라서, 본 발명은 상기 설명한 폴리뉴클레오티드에 의해 코딩되거나, 또는 상기 폴리펩티드의 생산 방법에 의해 얻을 수 있는 폴리펩티드를 포함한다. 본 발명의 다른 부분은 상기 설명한 폴리펩티드에 특이적으로 결합하는 항체를 포함한다. 본 발명의 추가의 실시양태는
a) 상기 설명한 숙주 세포 또는 파에실로미세스 디바리카투스 세포를 상기 숙주 세포에서 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나의 생산을 허용하는 조건 하에서 배양하는 단계; 및
b) 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나를 상기 숙주 세포 또는 파에실로미세스 디바리카투스 세포 또는 배양 배지로부터 얻는 단계
를 포함하는, 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I의 제조 방법, 또는 이들의 조합물의 제조 방법을 포함한다.
또한, 본 발명은 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및 카노클라빈 I의 군으로부터 선택되는 적어도 하나의 화합물의 제조를 위한, 상기 설명한 폴리뉴클레오티드, 또는 상기 설명한 벡터 또는 상기 설명한 숙주 세포 또는 파에실로미세스 디바리카투스 세포의 용도를 포함한다.
도 1은 시클로클라빈, 또는 경로의 다른 가지 상에서 아그로클라빈 및/또는 페스투클라빈을 생성하는 대사 경로의 상이한 단계의 개략도를 보여준다. 경로의 분기점은 카노클라빈 I이 합성된 후에 위치하고, 이어서 이것은 EasH, EasD, EasA 및 EasG 활성의 조합에 의해 추가로 변형되어 시클로클라빈을 생산하거나, 카노클라빈 I은 EasD 활성을 통해 카노클라빈 알데히드로 전환되고, 존재하는 EasA 활성에 따라 EasA 및 EasG 활성의 조합을 통해 아그로클라빈 및/또는 페스투클라빈으로 추가로 변형된다. EasA 활성은 아그로클라빈의 경우에 EasA 이소머라제 활성, 또는 페스투클라빈의 경우에 EasA 리덕타제 활성일 수 있다.
도 2는 카노클라빈 I으로부터 EasD, EasH, EasA 및 EasG 활성의 조합을 통해 시클로클라빈으로, 또는 카노클라빈으로부터 EasD 활성을 통해 카노클라빈 알데히드로, 및 카노클라빈 알데히드로부터 "EasA (Tyr176)"에 의해 매개된 EasG 및 EasA 리덕타제 활성의 조합을 통해 페스투클라빈으로, 또는 카노클라빈 알데히드로부터 "EasA (Phe176)"에 의해 매개된 EasG 및 EasA 이소머라제 활성의 조합을 통해 아그로클라빈으로 이르는 대사 경로의 단계에 관여된 상이한 단계 및 상이한 Eas 활성의 개략도를 보여준다. 도 2는 또한 클라빈-유형 알칼로이드를 생산하는 몇몇 예시적 천연 맥각 생산자 유기체를 거명한다. 상기 천연 맥각 생산자 유기체는 시클로클라빈의 경우에 아스페르길루스 자포니쿠스(Aspergillus japonicus) (에이. 자포니쿠스), 아그로클라빈의 경우에 클라비셉스 푸르푸레아(Claviceps purpurea) (씨. 푸르푸레아), 및 페스투클라빈의 경우에 아스페르길루스 푸미가투스(Aspergillus fumigatus) (에이. 푸미가투스)이다.
도 3a 내지 3d는 DmaW 활성을 갖는 아스페르길루스 자포니쿠스 폴리펩티드에 대한 몇몇 상동체 폴리펩티드의 아미노산 서열 정렬의 연속적인 파트를 도시한 것이다. 아미노산은 표준 단일 문자 아미노산 코드에 따라 표시된다. 상이한 서열의 명칭은 그로부터 이들 상동체 폴리펩티드가 얻어질 수 있는 유기체의 명칭, 및 이들 서열이 서열 목록에 나열되는 서열 번호를 포함한다. 아미노산 서열 정렬은 상기 군의 서열 내에서 또한 보존된, 덜 보존된 및 훨씬 덜 보존된 아미노산을 도시하고, 여기서, 흑색 음영표시의 아미노산 위치는 강한 보존, 즉, 100% 보존되는 것을 나타내고, 회색 음영표시의 아미노산 위치는 더 적은 보존을 나타내고, 백색 배경을 갖는 아미노산은 훨씬 덜 보존된 아미노산 위치를 나타낸다. 정렬 내의 아미노산 서열 내의 획은 특정 서열 내에 포함되지 않은 아미노산 위치를 나타낸다. 아미노산 위치의 군 위 또는 아래의 흑색 막대는 강한 서열 보존의 영역을 도시하고, 이것은 보존된 서열 모티프를 나타낸다. 정렬은 또한 각각의 아미노산 위치에서 고도로 보존된 아미노산을 확인하기 위해 상기 군의 유전자에 대한 컨센서스 서열 (서열 190)을 포함한다. 상기 아미노산 서열 정렬에 의해 제공된 보존된 및 덜 보존된 아미노산에 대한 정보는 아미노산 서열 정렬에 의해 구성되지 않은 아미노산 서열이 아스페르길루스 자포니쿠스 폴리펩티드의 진정한 상동체일 것인지 결정하기 위해 사용될 수 있다. 잠재적인 상동체 또는 자연 발생 또는 인공 생산된 돌연변이체의 아미노산 서열은 아마도 DmaW 활성을 가질 것이고, 보다 보존된 아미노산 위치는 문제의 아미노산 서열로 구성될 것이다.
도 4a 내지 4c는 EasF 활성을 갖는 아스페르길루스 자포니쿠스 폴리펩티드에 대한 몇몇 상동체 폴리펩티드의 아미노산 서열 정렬의 연속적인 파트를 도시한 것이다. 서열 명칭 및 기호, 예를 들어 흑색 또는 회색 음영표시 또는 컨센서스 서열 (서열 154)은 도 3a 내지 3d에 사용된 서열 명칭 및 기호에 대해 설명된 것과 동일한 의미를 갖는다.
도 5a 내지 5e는 EasE 활성을 갖는 아스페르길루스 자포니쿠스 폴리펩티드에 대한 몇몇 상동체 폴리펩티드의 아미노산 서열 정렬의 연속적인 파트를 도시한 것이다. 서열 명칭 및 기호, 예를 들어 흑색 또는 회색 음영표시, 또는 (서열 191)에 도시된 코어 컨센서스를 포함한 컨센서스 서열 (서열 185)은 도 3a 내지 3d에 사용된 서열 명칭 및 기호에 대해 설명된 것과 동일한 의미를 갖는다.
도 6a 내지 6d는 EasC 활성을 갖는 아스페르길루스 자포니쿠스 폴리펩티드에 대한 몇몇 상동체 폴리펩티드의 아미노산 서열 정렬의 연속적인 파트를 도시한 것이다. 서열 명칭 및 기호, 예를 들어 흑색 또는 회색 음영표시 또는 컨센서스 서열 (서열 151)은 도 3a 내지 3d에 사용된 서열 명칭 및 기호에 대해 설명된 것과 동일한 의미를 갖는다.
도 7a 내지 7b는 EasD 활성을 갖는 아스페르길루스 자포니쿠스 폴리펩티드에 대한 몇몇 상동체 폴리펩티드의 아미노산 서열 정렬의 연속적인 파트를 도시한 것이다. 서열 명칭 및 기호, 예를 들어 흑색 또는 회색 음영표시 또는 컨센서스 서열 (서열 152)는 도 3a 내지 3d에 사용된 서열 명칭 및 기호에 대해 설명된 것과 동일한 의미를 갖는다. 도 7a에서 4개의 흑색 화살표 및 도 7b에서 2개의 흑색 화살표는 EasD 활성을 갖는 단백질의 보조인자-결합 모티프 TGxxxGxG (서열 188)의 보존된 아미노산 및 활성 부위 모티프 YxxxK (서열 198)의 보존된 아미노산을 지시한다 (Kavanagh 2008, Cell Mol Life Sci 65:3895-3906).
도 8은 EasH 활성을 갖는 아스페르길루스 자포니쿠스 폴리펩티드에 대한 상동체 폴리펩티드의 아미노산 서열 정렬을 도시한 것이다. 서열 명칭 및 기호, 예를 들어 흑색 또는 회색 음영표시 또는 컨센서스 서열 (서열 183)은 도 3a 내지 3d에 사용된 서열 명칭 및 기호에 대해 설명된 것과 동일한 의미를 갖는다.
도 9a 내지 9d는 EasA 활성을 갖는 아스페르길루스 자포니쿠스 폴리펩티드에 대한 몇몇 상동체 폴리펩티드의 아미노산 서열 정렬의 연속적인 파트를 도시한 것이다. 서열 명칭 및 기호, 예를 들어 흑색 또는 회색 음영표시, 흑색 막대 또는 컨센서스 서열 (서열 184)는 도 3a 내지 3d에 사용된 서열 명칭 및 기호에 대해 설명된 것과 동일한 의미를 갖는다. 도 9b는 Y (아미노산 티로신을 나타냄) 또는 F (아미노산 페닐알라닌을 나타냄)를 포함하는 아미노산 위치를 지시하는 추가의 흑색 화살표를 포함한다. 상기 위치에서 Y를 갖는 아미노산 서열은 EasA 리덕타제 활성을 갖는 폴리펩티드를 나타내는 반면, 상기 위치에서 F를 갖는 아미노산 서열은 EasA 이소머라제 활성을 갖는 폴리펩티드를 나타낸다. 이들 활성은 때때로 EasA 리덕타제 및 EasA 이소머라제 활성에 대해 각각 EasA (Tyr176) 및 EasA (Phe176)로서도 또한 설명되고, 여기서 숫자 176은 각각 EasA 리덕타제 또는 EasA 이소머라제 활성을 갖는, 아스페르길루스 푸미가투스의 폴리펩티드 내에서 Y 또는 F를 갖는 상응하는 아미노산 위치를 의미한다 (Cheng et al.; 2010, J. AM. CHEM. SOC, 132: p12835-12837).
도 10a 내지 10c는 EasG 활성을 갖는 아스페르길루스 자포니쿠스 폴리펩티드에 대한 몇몇 상동체 폴리펩티드의 아미노산 서열 정렬의 연속적인 파트를 도시한 것이다. 서열 명칭 및 기호, 예를 들어 흑색 또는 회색 음영표시 또는 컨센서스 서열 (서열 183)은 도 3a 내지 3d에 사용된 서열 명칭 및 기호에 대해 설명된 것과 동일한 의미를 갖는다.
도 11은 상단부터 하단까지 효모 균주의 상청액의 크로마토그램을 보여준다: EYS1849: 총 이온 크로마토그램; EYS1849: m/z=239.1543+/-0.01 Da의 추출된 이온 크로마토그램; EYS1849: m/z=241.1699+/-0.01 Da의 추출된 이온 크로마토그램; EYS1851: 총 이온 크로마토그램; EYS1851: m/z=239.1543+/-0.01 Da의 추출된 이온 크로마토그램; EYS1851: m/z=241.1699+/-0.01 Da의 추출된 이온 크로마토그램.
도 12는 LC-MS에 의해 분석된 CEY2871 및 CEY3631의 상청액의 분석을 보여준다. 카노클라빈 I의 질량 (m/z = 257.164+/-0.01 Da)을 TIC (총 이온 크로마토그램)으로부터 추출하였고, 이것은 CEY3631 내에서 2.0 min+/-0.1 min의 체류 시간에서 피크를 보여주고, 이것은 CEY2871 내에서 부재하였다. 피크를 정제하고 NMR에 의해 분석하였다.
도 13은 LC-MS에 의해 분석한 균주 CEY3631 및 CEY3645의 상청액의 분석을 보여준다. 카노클라빈 I의 질량 (m/z = 257.164+/-0.01 Da)을 TIC (총 이온 크로마토그램)로부터 추출하였고, 이것은 CEY3631 및 CEY3645 내에서 체류 시간 2.0+/-0.1 min에서 피크를 보여준다. 피크 하 면적을 통합하였고 (삽입도), 이것은 EY3631에 비해 CEY3645 내에서 카노클라빈 I의 증가를 밝혔다.
도 14: 균주 CEY2557 및 CEY2625를 LC-MS에 의해 분석하였다. 피크는 두 균주 모두에서 7.8 min+/-0.2 min의 체류 시간에서 보였고, 이것은 Me-DMAT의 예측된 질량 (m/z = 287.175+/-0.01 Da)을 가졌다. 정체는 NMR에 의해 확인하였다. 피크 하 면적을 통합하였고, 이것은 CEY2557에 비해 CEY2625 내에서 Me-DMAT의 2배 초과의 증가를 밝혔다.
도 15: 균주 CEY2557 및 CEY2625를 LC-MS에 의해 분석하였다 (도 6 참조). 피크 하 면적을 통합하였고, 이것은 CEY2557에 비해 CEY2625 내에서 Me-DMAT의 2배 초과의 증가를 밝혔다. DMAT의 면적을 또한 도시한다.
도 16: 균주 CEY2557, CEY2563 및 CEY2575를 LC-MS에 의해 분석하였다. 모든 균주에서, 피크는 7.8 min+/-0.2 min의 체류 시간에서 보였고, 이것은 Me-DMAT의 예측된 질량 (m/z = 287.175+/-0.01 Da)을 가졌다. 피크 하 면적을 통합하였고, 이것은 3가지 모든 균주에서 유사한 수준의 화합물 (삽입도)을 보여준다. 화합물의 정체는 NMR에 의해 확인하였다.
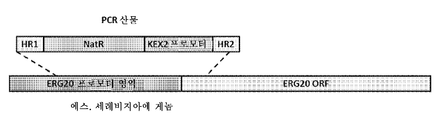
도 17: ERG20 유전자 내로 통합을 위해 사용된 PCR 산물, 및 효모 게놈 내의 표적 영역의 개략도이다. HR1 및 HR2는 게놈 서열에 상동성이고, NatR-ScKex2 프로모터 구축물의 상동성 재조합에 의한 통합을 허용하여, 천연 ERG20 프로모터를 파괴시킨다
도 18: 균주 EYS1538 및 EYS1926을 LC-MS에 의해 분석하였다. 피크 하 면적을 통합하였고, 이것은 EYS1538에 비해 EYS1926 내에서 Me-DMAT의 2배 초과의 증가를 밝혔다.
도 19: EYS2098 및 EYS2099로부터 배양 상청액의 분석은 이들 균주에 의해 생산된 카노클라빈 I 및 시클로클라빈의 상대적인 양 (곡선 하 면적)을 보여준다.
도 20a 및 20b: EYS2055 및 EYS2056에서 DMAT 및 Me-DMAT의 생산. 둘 모두 Me-DMAT에 대해 2-단계 이종 경로를 발현한다. 또한, EYS2056은 여분의, 구성적으로 발현된 카피의 ScIdi1을 함유한다.
도 21: EYS1934 (대조군) 및 EYS2206 (EasC) 내에서 시클로클라빈 (CCL) 및 페스투클라빈 (MW241)의 생산을 분석하였고, 이것은 추가의 카피의 유전자 AjEasC의 발현 때문에 EYS2206 내에서 증가된 생산을 보여주었다.
도 22: EYS2124 (에이. 자포니쿠스로부터 EasE) 및 EYS2125 (엔. 롤리이로부터 EasE)로부터 배양 상청액의 분석은 이들 균주에 의해 생산된 시클로클라빈 및 페스투클라빈 (MW241)의 상대적인 양 (곡선 하 면적)을 보여준다.
도 23: 각각 상이한 DNA 코돈 용법을 갖는 EasE 유전자를 갖지만 모두 에이. 자포니쿠스로부터 상응하는 효소를 코딩하는, 전장 시클로클라빈 경로를 갖는 효모 균주로부터 배양 상청액의 분석. 상이한 코돈 사용은 이들 균주에 의해 생산되는 다양한 양 (곡선 하 면적으로서 도시됨)의 시클로클라빈 및 페스투클라빈 (MW241)을 생성시킨다.
도 24: EYS2006을 글리세롤 없이 (대조군) 또는 각각 2.5%, 5%, 및 10% 글리세롤과 함께 성장시켰다. 시클로클라빈 및 페스투클라빈 (MW241)의 생산은 증가하는 농도의 글리세롤과 함께 증가하였다.
도 25: EYS2209에 비해 EYS1934 (좌측), 및 EYS2297에 비해 EYS2209 내에서 시클로클라빈의 생산.
도 26: 유가식 발효에서 CCL 및 FCL의 생산. 화합물의 농도 (mg/L), 및 측정된 OD600을 발효의 지속 시간에 대해 보여준다.
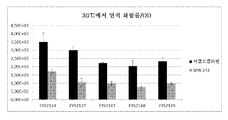
도 27: 상이한 유전적 배경을 갖는 7개의 효모 균주 (사카로미세스 세레비지아에(Saccharomyces cerevisiae)) 내에서 CCL 및 FCL의 생산 (표 58 참조).
도 2는 카노클라빈 I으로부터 EasD, EasH, EasA 및 EasG 활성의 조합을 통해 시클로클라빈으로, 또는 카노클라빈으로부터 EasD 활성을 통해 카노클라빈 알데히드로, 및 카노클라빈 알데히드로부터 "EasA (Tyr176)"에 의해 매개된 EasG 및 EasA 리덕타제 활성의 조합을 통해 페스투클라빈으로, 또는 카노클라빈 알데히드로부터 "EasA (Phe176)"에 의해 매개된 EasG 및 EasA 이소머라제 활성의 조합을 통해 아그로클라빈으로 이르는 대사 경로의 단계에 관여된 상이한 단계 및 상이한 Eas 활성의 개략도를 보여준다. 도 2는 또한 클라빈-유형 알칼로이드를 생산하는 몇몇 예시적 천연 맥각 생산자 유기체를 거명한다. 상기 천연 맥각 생산자 유기체는 시클로클라빈의 경우에 아스페르길루스 자포니쿠스(Aspergillus japonicus) (에이. 자포니쿠스), 아그로클라빈의 경우에 클라비셉스 푸르푸레아(Claviceps purpurea) (씨. 푸르푸레아), 및 페스투클라빈의 경우에 아스페르길루스 푸미가투스(Aspergillus fumigatus) (에이. 푸미가투스)이다.
도 3a 내지 3d는 DmaW 활성을 갖는 아스페르길루스 자포니쿠스 폴리펩티드에 대한 몇몇 상동체 폴리펩티드의 아미노산 서열 정렬의 연속적인 파트를 도시한 것이다. 아미노산은 표준 단일 문자 아미노산 코드에 따라 표시된다. 상이한 서열의 명칭은 그로부터 이들 상동체 폴리펩티드가 얻어질 수 있는 유기체의 명칭, 및 이들 서열이 서열 목록에 나열되는 서열 번호를 포함한다. 아미노산 서열 정렬은 상기 군의 서열 내에서 또한 보존된, 덜 보존된 및 훨씬 덜 보존된 아미노산을 도시하고, 여기서, 흑색 음영표시의 아미노산 위치는 강한 보존, 즉, 100% 보존되는 것을 나타내고, 회색 음영표시의 아미노산 위치는 더 적은 보존을 나타내고, 백색 배경을 갖는 아미노산은 훨씬 덜 보존된 아미노산 위치를 나타낸다. 정렬 내의 아미노산 서열 내의 획은 특정 서열 내에 포함되지 않은 아미노산 위치를 나타낸다. 아미노산 위치의 군 위 또는 아래의 흑색 막대는 강한 서열 보존의 영역을 도시하고, 이것은 보존된 서열 모티프를 나타낸다. 정렬은 또한 각각의 아미노산 위치에서 고도로 보존된 아미노산을 확인하기 위해 상기 군의 유전자에 대한 컨센서스 서열 (서열 190)을 포함한다. 상기 아미노산 서열 정렬에 의해 제공된 보존된 및 덜 보존된 아미노산에 대한 정보는 아미노산 서열 정렬에 의해 구성되지 않은 아미노산 서열이 아스페르길루스 자포니쿠스 폴리펩티드의 진정한 상동체일 것인지 결정하기 위해 사용될 수 있다. 잠재적인 상동체 또는 자연 발생 또는 인공 생산된 돌연변이체의 아미노산 서열은 아마도 DmaW 활성을 가질 것이고, 보다 보존된 아미노산 위치는 문제의 아미노산 서열로 구성될 것이다.
도 4a 내지 4c는 EasF 활성을 갖는 아스페르길루스 자포니쿠스 폴리펩티드에 대한 몇몇 상동체 폴리펩티드의 아미노산 서열 정렬의 연속적인 파트를 도시한 것이다. 서열 명칭 및 기호, 예를 들어 흑색 또는 회색 음영표시 또는 컨센서스 서열 (서열 154)은 도 3a 내지 3d에 사용된 서열 명칭 및 기호에 대해 설명된 것과 동일한 의미를 갖는다.
도 5a 내지 5e는 EasE 활성을 갖는 아스페르길루스 자포니쿠스 폴리펩티드에 대한 몇몇 상동체 폴리펩티드의 아미노산 서열 정렬의 연속적인 파트를 도시한 것이다. 서열 명칭 및 기호, 예를 들어 흑색 또는 회색 음영표시, 또는 (서열 191)에 도시된 코어 컨센서스를 포함한 컨센서스 서열 (서열 185)은 도 3a 내지 3d에 사용된 서열 명칭 및 기호에 대해 설명된 것과 동일한 의미를 갖는다.
도 6a 내지 6d는 EasC 활성을 갖는 아스페르길루스 자포니쿠스 폴리펩티드에 대한 몇몇 상동체 폴리펩티드의 아미노산 서열 정렬의 연속적인 파트를 도시한 것이다. 서열 명칭 및 기호, 예를 들어 흑색 또는 회색 음영표시 또는 컨센서스 서열 (서열 151)은 도 3a 내지 3d에 사용된 서열 명칭 및 기호에 대해 설명된 것과 동일한 의미를 갖는다.
도 7a 내지 7b는 EasD 활성을 갖는 아스페르길루스 자포니쿠스 폴리펩티드에 대한 몇몇 상동체 폴리펩티드의 아미노산 서열 정렬의 연속적인 파트를 도시한 것이다. 서열 명칭 및 기호, 예를 들어 흑색 또는 회색 음영표시 또는 컨센서스 서열 (서열 152)는 도 3a 내지 3d에 사용된 서열 명칭 및 기호에 대해 설명된 것과 동일한 의미를 갖는다. 도 7a에서 4개의 흑색 화살표 및 도 7b에서 2개의 흑색 화살표는 EasD 활성을 갖는 단백질의 보조인자-결합 모티프 TGxxxGxG (서열 188)의 보존된 아미노산 및 활성 부위 모티프 YxxxK (서열 198)의 보존된 아미노산을 지시한다 (Kavanagh 2008, Cell Mol Life Sci 65:3895-3906).
도 8은 EasH 활성을 갖는 아스페르길루스 자포니쿠스 폴리펩티드에 대한 상동체 폴리펩티드의 아미노산 서열 정렬을 도시한 것이다. 서열 명칭 및 기호, 예를 들어 흑색 또는 회색 음영표시 또는 컨센서스 서열 (서열 183)은 도 3a 내지 3d에 사용된 서열 명칭 및 기호에 대해 설명된 것과 동일한 의미를 갖는다.
도 9a 내지 9d는 EasA 활성을 갖는 아스페르길루스 자포니쿠스 폴리펩티드에 대한 몇몇 상동체 폴리펩티드의 아미노산 서열 정렬의 연속적인 파트를 도시한 것이다. 서열 명칭 및 기호, 예를 들어 흑색 또는 회색 음영표시, 흑색 막대 또는 컨센서스 서열 (서열 184)는 도 3a 내지 3d에 사용된 서열 명칭 및 기호에 대해 설명된 것과 동일한 의미를 갖는다. 도 9b는 Y (아미노산 티로신을 나타냄) 또는 F (아미노산 페닐알라닌을 나타냄)를 포함하는 아미노산 위치를 지시하는 추가의 흑색 화살표를 포함한다. 상기 위치에서 Y를 갖는 아미노산 서열은 EasA 리덕타제 활성을 갖는 폴리펩티드를 나타내는 반면, 상기 위치에서 F를 갖는 아미노산 서열은 EasA 이소머라제 활성을 갖는 폴리펩티드를 나타낸다. 이들 활성은 때때로 EasA 리덕타제 및 EasA 이소머라제 활성에 대해 각각 EasA (Tyr176) 및 EasA (Phe176)로서도 또한 설명되고, 여기서 숫자 176은 각각 EasA 리덕타제 또는 EasA 이소머라제 활성을 갖는, 아스페르길루스 푸미가투스의 폴리펩티드 내에서 Y 또는 F를 갖는 상응하는 아미노산 위치를 의미한다 (Cheng et al.; 2010, J. AM. CHEM. SOC, 132: p12835-12837).
도 10a 내지 10c는 EasG 활성을 갖는 아스페르길루스 자포니쿠스 폴리펩티드에 대한 몇몇 상동체 폴리펩티드의 아미노산 서열 정렬의 연속적인 파트를 도시한 것이다. 서열 명칭 및 기호, 예를 들어 흑색 또는 회색 음영표시 또는 컨센서스 서열 (서열 183)은 도 3a 내지 3d에 사용된 서열 명칭 및 기호에 대해 설명된 것과 동일한 의미를 갖는다.
도 11은 상단부터 하단까지 효모 균주의 상청액의 크로마토그램을 보여준다: EYS1849: 총 이온 크로마토그램; EYS1849: m/z=239.1543+/-0.01 Da의 추출된 이온 크로마토그램; EYS1849: m/z=241.1699+/-0.01 Da의 추출된 이온 크로마토그램; EYS1851: 총 이온 크로마토그램; EYS1851: m/z=239.1543+/-0.01 Da의 추출된 이온 크로마토그램; EYS1851: m/z=241.1699+/-0.01 Da의 추출된 이온 크로마토그램.
도 12는 LC-MS에 의해 분석된 CEY2871 및 CEY3631의 상청액의 분석을 보여준다. 카노클라빈 I의 질량 (m/z = 257.164+/-0.01 Da)을 TIC (총 이온 크로마토그램)으로부터 추출하였고, 이것은 CEY3631 내에서 2.0 min+/-0.1 min의 체류 시간에서 피크를 보여주고, 이것은 CEY2871 내에서 부재하였다. 피크를 정제하고 NMR에 의해 분석하였다.
도 13은 LC-MS에 의해 분석한 균주 CEY3631 및 CEY3645의 상청액의 분석을 보여준다. 카노클라빈 I의 질량 (m/z = 257.164+/-0.01 Da)을 TIC (총 이온 크로마토그램)로부터 추출하였고, 이것은 CEY3631 및 CEY3645 내에서 체류 시간 2.0+/-0.1 min에서 피크를 보여준다. 피크 하 면적을 통합하였고 (삽입도), 이것은 EY3631에 비해 CEY3645 내에서 카노클라빈 I의 증가를 밝혔다.
도 14: 균주 CEY2557 및 CEY2625를 LC-MS에 의해 분석하였다. 피크는 두 균주 모두에서 7.8 min+/-0.2 min의 체류 시간에서 보였고, 이것은 Me-DMAT의 예측된 질량 (m/z = 287.175+/-0.01 Da)을 가졌다. 정체는 NMR에 의해 확인하였다. 피크 하 면적을 통합하였고, 이것은 CEY2557에 비해 CEY2625 내에서 Me-DMAT의 2배 초과의 증가를 밝혔다.
도 15: 균주 CEY2557 및 CEY2625를 LC-MS에 의해 분석하였다 (도 6 참조). 피크 하 면적을 통합하였고, 이것은 CEY2557에 비해 CEY2625 내에서 Me-DMAT의 2배 초과의 증가를 밝혔다. DMAT의 면적을 또한 도시한다.
도 16: 균주 CEY2557, CEY2563 및 CEY2575를 LC-MS에 의해 분석하였다. 모든 균주에서, 피크는 7.8 min+/-0.2 min의 체류 시간에서 보였고, 이것은 Me-DMAT의 예측된 질량 (m/z = 287.175+/-0.01 Da)을 가졌다. 피크 하 면적을 통합하였고, 이것은 3가지 모든 균주에서 유사한 수준의 화합물 (삽입도)을 보여준다. 화합물의 정체는 NMR에 의해 확인하였다.
도 17: ERG20 유전자 내로 통합을 위해 사용된 PCR 산물, 및 효모 게놈 내의 표적 영역의 개략도이다. HR1 및 HR2는 게놈 서열에 상동성이고, NatR-ScKex2 프로모터 구축물의 상동성 재조합에 의한 통합을 허용하여, 천연 ERG20 프로모터를 파괴시킨다
도 18: 균주 EYS1538 및 EYS1926을 LC-MS에 의해 분석하였다. 피크 하 면적을 통합하였고, 이것은 EYS1538에 비해 EYS1926 내에서 Me-DMAT의 2배 초과의 증가를 밝혔다.
도 19: EYS2098 및 EYS2099로부터 배양 상청액의 분석은 이들 균주에 의해 생산된 카노클라빈 I 및 시클로클라빈의 상대적인 양 (곡선 하 면적)을 보여준다.
도 20a 및 20b: EYS2055 및 EYS2056에서 DMAT 및 Me-DMAT의 생산. 둘 모두 Me-DMAT에 대해 2-단계 이종 경로를 발현한다. 또한, EYS2056은 여분의, 구성적으로 발현된 카피의 ScIdi1을 함유한다.
도 21: EYS1934 (대조군) 및 EYS2206 (EasC) 내에서 시클로클라빈 (CCL) 및 페스투클라빈 (MW241)의 생산을 분석하였고, 이것은 추가의 카피의 유전자 AjEasC의 발현 때문에 EYS2206 내에서 증가된 생산을 보여주었다.
도 22: EYS2124 (에이. 자포니쿠스로부터 EasE) 및 EYS2125 (엔. 롤리이로부터 EasE)로부터 배양 상청액의 분석은 이들 균주에 의해 생산된 시클로클라빈 및 페스투클라빈 (MW241)의 상대적인 양 (곡선 하 면적)을 보여준다.
도 23: 각각 상이한 DNA 코돈 용법을 갖는 EasE 유전자를 갖지만 모두 에이. 자포니쿠스로부터 상응하는 효소를 코딩하는, 전장 시클로클라빈 경로를 갖는 효모 균주로부터 배양 상청액의 분석. 상이한 코돈 사용은 이들 균주에 의해 생산되는 다양한 양 (곡선 하 면적으로서 도시됨)의 시클로클라빈 및 페스투클라빈 (MW241)을 생성시킨다.
도 24: EYS2006을 글리세롤 없이 (대조군) 또는 각각 2.5%, 5%, 및 10% 글리세롤과 함께 성장시켰다. 시클로클라빈 및 페스투클라빈 (MW241)의 생산은 증가하는 농도의 글리세롤과 함께 증가하였다.
도 25: EYS2209에 비해 EYS1934 (좌측), 및 EYS2297에 비해 EYS2209 내에서 시클로클라빈의 생산.
도 26: 유가식 발효에서 CCL 및 FCL의 생산. 화합물의 농도 (mg/L), 및 측정된 OD600을 발효의 지속 시간에 대해 보여준다.
도 27: 상이한 유전적 배경을 갖는 7개의 효모 균주 (사카로미세스 세레비지아에(Saccharomyces cerevisiae)) 내에서 CCL 및 FCL의 생산 (표 58 참조).
일반적인 정의
용어 "시클로클라빈"은 하기 화학식 I의 화합물을 의미한다.
<화학식 I>
용어 "페스투클라빈"은 하기 화학식 II의 화합물을 의미한다.
<화학식 II>
용어 "아그로클라빈"은 하기 화학식 III의 화합물을 의미한다.
<화학식 III>
용어 "카노클라빈 알데히드"는 하기 화학식 IV의 화합물을 의미한다.
<화학식 IV>
용어 "카노클라빈 I"은 하기 화학식 V의 화합물을 의미한다.
<화학식 V>
용어 "ME-DMAT"는 하기 화학식 VI의 화합물을 의미한다.
<화학식 VI>
용어 "DMAT"는 하기 화학식 VII의 화합물을 의미한다.
<화학식 VII>
용어 "DMAPP"는 하기 화학식 VIII의 화합물을 의미한다.
<화학식 VIII>
용어 "트립토판"은 아미노산 트립토판, 바람직하게는 L-트립토판, 또는 그의 염 중의 하나를 의미한다.
용어 "IPP"는 화학식 IX의 화합물인 이소펜테닐 피로인산염 또는 그의 염 중의 하나를 의미한다.
용어 "DmaW 활성"은 서열 1의 폴리펩티드와 동일한 또는 유사한 기능을 충족하는, 즉, 디메틸알릴트립토판 신타제 활성을 갖고 위치 C4에서 L-트립토판의 프레닐화를 촉매하여 4-디메틸알릴트립토판 ((S)-4-(3-메틸-2-부테닐)-트립토판, 또는 DMAT)의 형성을 야기하는, 폴리펩티드 또는 상기 폴리펩티드를 포함하는 미생물의 능력을 의미한다.
용어 "EasF 활성"은 서열 6의 폴리펩티드와 동일한 또는 유사한 기능을 충족하는, 즉, N-메틸트랜스퍼라제 활성을 갖고 DMAT의 Me-DMAT로의 메틸화를 촉매하는, 폴리펩티드 또는 상기 폴리펩티드를 포함하는 미생물의 능력을 의미한다.
용어 "EasE 활성"은 서열 5의 폴리펩티드와 동일한 또는 유사한 기능을 충족하는, 즉, EasC 활성이 또한 존재하는 경우에 Me-DMAT의 카노클라빈 I으로의 전환을 촉매하는, 폴리펩티드 또는 상기 폴리펩티드를 포함하는 미생물의 능력을 의미한다.
용어 "EasC 활성"은 서열 3의 폴리펩티드와 동일한 또는 유사한 기능을 충족하는, 즉, EasE 활성이 또한 존재하는 경우에 Me-DMAT의 카노클라빈 I으로의 전환을 촉매하는, 폴리펩티드 또는 상기 폴리펩티드를 포함하는 미생물의 능력을 의미한다.
용어 "EasD 활성"은 서열 4의 폴리펩티드와 동일한 또는 유사한 기능을 충족하는, 즉, 옥시도리덕타제 활성을 갖고 카노클라빈 I의 카노클라빈 알데히드로의 전환을 촉매하는, 폴리펩티드 또는 상기 폴리펩티드를 포함하는 미생물의 능력을 의미한다.
용어 "EasH 활성"은 서열 8의 폴리펩티드와 동일한 또는 유사한 기능을 충족하는, 즉, EasD, EasA 및 EasG 활성이 또한 존재하는 경우에 카노클라빈 I의 시클로클라빈으로의 전환을 촉매하는, 폴리펩티드 또는 상기 폴리펩티드를 포함하는 미생물의 능력을 의미한다.
용어 "EasA 리덕타제 활성"은 서열 2의 폴리펩티드와 동일한 또는 유사한 기능을 충족하는, 즉, 리덕타제 활성을 갖고 EasG 활성이 존재하는 경우에 카노클라빈 알데히드의 페스투클라빈으로의 전환을 촉매하는, 폴리펩티드 또는 상기 폴리펩티드를 포함하는 미생물의 능력을 의미한다.
용어 "EasA 이소머라제 활성"은 서열 37의 폴리펩티드 (씨. 푸르푸레아 EasA)와 동일한 또는 유사한 기능을 충족하는, 즉, 이소머라제 활성을 갖고 EasG 활성이 존재하면 카노클라빈 알데히드의 아그로클라빈으로의 전환을 촉매하는, 폴리펩티드 또는 상기 폴리펩티드를 포함하는 미생물의 능력을 의미한다.
용어 "EasA 활성"은 예를 들어 서열 149의 위치 18에 Y의 F로의 돌연변이를 포함하는 서열 33의 네오티포디움 롤리이(Neotyphodium lolii)의 폴리펩티드처럼, 서열 2 또는 37의 폴리펩티드와 동일한 또는 유사한 기능을 충족하는, 즉, EasA 리덕타제 또는 EasA 이소머라제 활성을 갖거나 또는 두 활성을 모두 갖는, 폴리펩티드 또는 상기 폴리펩티드를 포함하는 미생물의 능력을 의미한다.
용어 "EasG 활성"은 서열 7의 폴리펩티드와 동일한 또는 유사한 기능을 충족하는, 즉, 옥시도리덕타제 활성을 갖고 존재하는 EasA 활성에 따라 카노클라빈 알데히드의 아그로클라빈 및/또는 페스투클라빈으로의 전환을 촉매하는, 및/또는 EasA, EasD 및 EasH 활성이 또한 존재할 경우 카노클라빈 I의 시클로클라빈으로의 전환을 촉매하는, 폴리펩티드 또는 상기 폴리펩티드를 포함하는 미생물의 능력을 의미한다.
용어 "ERG9 활성"은 2개의 파르네실 피로인산염 (FPP) 모이어티를 연결하여 스테롤 생합성 경로에서 스쿠알렌을 형성하는, 폴리펩티드 또는 상기 폴리펩티드를 포함하는 미생물의 능력을 의미한다. ERG9 활성은 대체로 스쿠알렌 신타제에 의헤 제공된다.
용어 "ERG20 활성"은 이소프레노이드 및 스테롤 생합성을 위한 C15 파르네실 피로인산염 단위의 형성을 촉매하는, 폴리펩티드 또는 상기 폴리펩티드를 포함하는 미생물의 능력을 의미한다.
용어 "HMG-CoA 리덕타제 활성"은 HMG-CoA의 메발로네이트로의 전환을 촉매하는, 폴리펩티드 또는 상기 폴리펩티드를 포함하는 미생물의 능력을 의미한다.
용어 "약"은 대체로, 대략, 쯤, 또는 가까이에를 의미하기 위해 본원에서 사용된다. 용어 "약"이 수치 범위와 함께 사용될 때, 이것은 경계를 제시된 수치 값 위 및 아래로 확장함으로써 그 범위를 변경한다. 일반적으로, 용어 "약"은 수치 값을 언급된 값의 20%, 바람직하게는 15%, 보다 바람직하게는 10%, 가장 바람직하게는 5%의 변동만큼 위 및 아래로 (더 높게 또는 더 낮게) 변경하기 위해 본원에서 사용된다.
용어 "게놈" 또는 "게놈 DNA"는 숙주 유기체의 유전가능한 유전 정보를 의미한다. 상기 게놈 DNA는 핵의 DNA (염색체 DNA), 염색체외 DNA, 및 소기관 DNA (예를 들어, 미토콘드리아의)를 포함하는 세포 또는 유기체의 전체 유전 물질을 포함한다. 바람직하게는, 용어 게놈 또는 게놈 DNA는 핵의 염색체 DNA를 의미한다.
용어 "염색체 DNA" 또는 "염색체 DNA 서열"은 세포 주기 상태와 무관한 세포 핵의 게놈 DNA로서 이해되어야 한다. 염색체 DNA는 따라서 염색체 또는 염색질 내에 조직화될 수 있고, 압축되거나 또는 감기지 않을 수 있다. 염색체 DNA 내로의 삽입은 예를 들어, 중합효소 연쇄 반응 (PCR) 분석, 서던 블롯 분석, 형광 계내 혼성화 (FISH), 계내 PCR 및 차세대 서열결정 (NGS)처럼 관련 기술 분야에 알려진 다양한 방법에 의해 입증되고 분석될 수 있다.
용어 "프로모터"는 mRNA를 생산하기 위해 구조 유전자의 전사를 지시하는 폴리뉴클레오티드를 의미한다. 일반적으로, 프로모터는 구조 유전자의 출발 코돈에 근접하게 유전자의 5' 영역에 위치한다. 프로모터가 유도가능 프로모터일 경우, 전사 속도는 유도제에 반응하여 증가한다. 이와 대조적으로, 전사 속도는 프로모터가 구성적 프로모터이면 유도제에 의해 조절되지 않는다.
용어 "인핸서"는 폴리뉴클레오티드를 나타낸다. 인핸서는 전사 출발 부위에 대한 인핸서의 거리 또는 배향과 무관하게 특정 유전자가 mRNA로 전사되는 효율을 증가시킬 수 있다. 대체로, 인핸서는 프로모터, 5'-비번역 서열에 근접하게 또는 인트론 내에 위치한다.
폴리뉴클레오티드 서열은 그 서열이 외래 종으로부터 기원하거나, 또는 동일한 종으로부터 기원하지만 그의 본래 형태로부터 변형될 경우, 유기체 또는 제2 폴리뉴클레오티드 서열에 "이종성"이다. 예를 들어, 이종성 코딩 서열에 작동가능하게 연결된 프로모터는 프로모터가 유래되는 것과 상이한 종으로부터 유래하는 코딩 서열 또는 동일한 종으로부터 유래하지만 프로모터와 천연적으로 회합하지 않는 코딩 서열 (예를 들어 유전자 조작된 코딩 서열 또는 상이한 생태형 또는 변종으로부터의 대립유전자)을 의미한다.
"트랜스진", "트랜스제닉" 또는 "재조합"은 인간에 의해 조작된 폴리뉴클레오티드 또는 인간에 의해 조작된 폴리뉴클레오티드의 카피 또는 상보체를 의미한다. 예를 들어, 제2 폴리뉴클레오티드에 작동가능하게 연결된 프로모터를 포함하는 트랜스제닉 발현 카세트는 발현 카세트를 포함하는 단리된 핵산의 인간에 의한 조작 (예를 들어, 문헌 [Sambrook et al., Molecular Cloning-A Laboratory Manual, Cold Spring Harbor Laboratory, Cold Spring Harbor, New York, (1989)] 또는 [Current Protocols in Molecular Biology Volumes 1-3, John Wiley & Sons, Inc. (1994-1998)]에 기재된 방법에 의해)의 결과로서 제2 폴리뉴클레오티드에 이종성인 프로모터를 포함할 수 있다. 또 다른 예에서, 재조합 발현 카세트는 폴리뉴클레오티드가 자연에서 발견될 가능성이 극히 낮은 방식으로 조합된 폴리뉴클레오티드를 포함할 수 있다. 예를 들어, 인간에 의해 조작된 제한 부위 또는 플라스미드 벡터 서열은 제2 폴리뉴클레오티드의 측면에 인접하거나 프로모터를 제2 폴리뉴클레오티드로부터 분리할 수 있다. 통상의 기술자는 폴리뉴클레오티드가 많은 방식으로 조작될 수 있고 상기 예로 제한되지 않음을 알 것이다. 용어 "재조합"이 유기체 또는 세포, 예를 들어 미생물을 구체화하기 위해 사용되는 경우에, 이것은 유기체 또는 세포가 대체로 추후에 구체화되는 적어도 하나의 "트랜스진", "트랜스제닉" 또는 "재조합" 폴리뉴클레오티드를 포함함을 표현하기 위해 사용된다.
개개의 유기체에 "외인성"인 폴리뉴클레오티드는 유성 교배 이외의 다른 임의의 수단에 의해 유기체 내로 도입되는 폴리뉴클레오티드이다.
용어 "작동가능한 연결" 또는 "작동가능하게 연결된"은 일반적으로 유전자 제어 서열, 예를 들어 프로모터, 인핸서 또는 종결인자가, 그가 작동가능하게 연결되는 폴리뉴클레오티드, 예를 들어 폴리펩티드를 코딩하는 폴리뉴클레오티드에 대해 그의 기능을 발휘할 수 있는 배열을 의미하는 것으로서 이해된다. 상기 맥락에서, 기능은 예를 들어 핵산 서열의 발현, 즉, 전사 및/또는 번역의 제어를 의미할 수 있다. 상기 맥락에서, 제어는 예를 들어 발현, 즉, 전사 및, 적절한 경우 번역의 개시, 증가, 조절 또는 억제를 포함한다. 다시, 제어는 예를 들어, 조직- 및/또는 시간-특이적일 수 있다. 또한, 제어는 예를 들어 특정 화학물질, 스트레스, 병원체 등에 의해 유도가능한 것일 수 있다. 바람직하게는, 작동가능한 연결은 예를 들어 핵산 서열이 발현될 때 각각의 조절 요소가 그의 기능을 수행할 수 있도록 프로모터, 발현되는 핵산 서열, 및 적절한 경우 추가의 조절 요소, 예컨대, 예를 들어, 종결인자의 순차적인 배열을 의미하는 것으로 이해된다. 작동가능한 연결은 화학적 의미에서 반드시 직접적인 연결을 필요로 하지는 않는다. 예를 들어, 인핸서 서열과 같은 유전자 제어 서열은 또한 작동가능하게 연결된 폴리뉴클레오티드에 일정 거리에 존재하는 위치로부터 표적 서열에 대한 그의 기능을 발휘할 수 있다. 바람직한 배열은 발현되는 핵산 서열이 2개의 서열이 서로 공유 연결되도록 프로모터로서 기능하는 서열 다음에 위치하는 것이다. 프로모터 서열과 발현 카세트 내의 핵산 서열 사이의 거리는 바람직하게는 200개 염기쌍 미만, 특히 바람직하게는 100개 염기쌍 미만, 매우 특히 바람직하게는 50개 염기쌍 미만이다. 통상의 기술자는 상기 발현 카세트를 얻기 위한 다양한 방식에 친숙하다. 그러나, 발현 카세트는 또한 발현되는 핵산 서열이 예를 들어 상동성 재조합에 의해 또는 무작위 삽입에 의해 내인성 유전자 제어 요소, 예를 들어 내인성 프로모터의 제어 하에 존재하는 방식으로 구축될 수 있다. 상기 구축물도 본 발명의 목적을 위한 발현 카세트로서 이해된다.
용어 "발현 카세트"는 발현되는 폴리뉴클레오티드 서열이 그의 발현 (즉, 전사 및/또는 번역)을 가능하게 하거나 조절하는 적어도 하나의 유전자 제어 요소에 작동가능하게 연결된 구축물을 의미한다. 발현은 예를 들어 안정한 또는 일시적인, 구성적 또는 유도성 발현일 수 있다.
용어 "발현하다", "발현하는", "발현된" 및 "발현"은 코딩되는 단백질의 생성되는 효소 활성, 또는 언급되는 본원에서 규정되고 설명된 경로 또는 반응이 상기 유전자/경로가 발현되는 유기체에서 상기 경로 또는 반응을 통한 대사 흐름을 허용하는 수준에서 유전자 산물 (예를 들어, 본원에서 규정되고 설명되는 경로 또는 반응의 유전자의 생합성 효소)의 발현을 의미한다. 발현은 출발 유기체로서 사용하는 미생물의 유전자 변경에 의해 수행될 수 있다. 몇몇 실시양태에서, 미생물은 출발 미생물에 의해 또는 변경되지 않은 대등한 미생물에서 생산된 것에 비해 증가된 수준에서 유전자 산물을 발현하도록 유전적으로 변경 (예를 들어, 유전자 조작)될 수 있다. 유전자 변경은 특정 유전자의 발현과 연관된 조절 서열 또는 부위의 변경 또는 변형 (예를 들어 강한 프로모터, 유도가능 프로모터 또는 다중 프로모터의 부가에 의한 또는 발현이 구성적이도록 조절 서열의 제거에 의한), 특정 유전자의 염색체 위치의 변경, 특정 유전자, 예컨대 리보솜 결합 부위 또는 전사 종결인자에 인접한 핵산 서열의 변경, 특정 유전자의 카피수의 증가, 특정 유전자의 전사 및/또는 특정 유전자 산물의 번역에 연관되는 단백질 (예를 들어, 조절 단백질, 서프레서, 인핸서, 전사 활성제 등)의 변형, 또는 관련 기술 분야에 통상적인 기술을 사용하여 특정 유전자의 발현을 탈조절하는 임의의 다른 통상적인 수단 (예를 들어, 리프레서 단백질의 발현을 차단하기 위한 안티센스 핵산 분자의 사용을 포함하고 이로 제한되지 않음)을 포함하고 이로 제한되지 않는다.
몇몇 실시양태에서, 미생물은 비변경된 미생물에서의 유전자 산물의 발현 수준에 비해 증가되거나 더 낮은 수준에서 유전자 산물을 발현하도록 물리적으로 또는 환경적으로 변경될 수 있다. 예를 들어, 미생물은 전사 및/또는 번역이 향상되거나 증가되도록 특정 유전자의 전사 및/또는 특정 유전자 산물의 번역을 증가시키는 것으로 알려지거나 의심되는 물질로 처리되거나 이 물질의 존재 하에 배양될 수 있다. 대안적으로, 미생물은 전사 및/또는 번역이 향상되거나 증가되도록 특정 유전자의 전사 및/또는 특정 유전자 산물의 번역을 증가시키기 위해 선택된 온도에서 배양될 수 있다.
용어 "탈조절하다", "탈조절된" 및 "탈조절"은 미생물에서 적어도 하나의 유전자의 변경 또는 변형을 의미하고, 여기서 변경 또는 변형은 변경 또는 변형의 부재 하에서의 생산에 비해 미생물에서 제시된 화합물의 생산 효율을 증가시킨다. 몇몇 실시양태에서, 변경 또는 변형된 유전자는 미생물에서 생합성 효소의 수준 또는 활성이 변경 또는 변형되도록, 또는 수송 특이성 또는 효율이 변경 또는 변형되도록 생합성 경로의 효소, 또는 수송 단백질을 코딩한다. 몇몇 실시양태에서, 생합성 경로의 효소, 즉, 생합성 경로의 특이적 활성, 예를 들어 EasH 활성을 유도하는 폴리펩티드를 코딩하는 적어도 하나의 유전자는 효소의 수준 또는 활성이 비변경된 또는 야생형 유전자의 존재 하의 수준에 비해 향상되거나 증가되도록 변경 또는 변형된다. 탈조절은 또한 예를 들어 피드백 저항성이거나 또는 더 높거나 더 낮은 특이적 활성을 갖는 효소를 생성하기 위해 하나 이상의 유전자의 코딩 영역을 변경하는 것을 포함한다. 또한, 탈조절은 효소 또는 수송 단백질을 코딩하는 유전자의 발현을 조절하는 전사 인자 (예를 들어, 활성제, 리프레서)를 코딩하는 유전자의 유전자 변경을 추가로 포함한다. 용어 "탈조절하다", "탈조절된" 및 "탈조절"은 존재하는 탈조절의 유형에 대해 추가로 구체화될 수 있다. 특정 활성, 예를 들어 EasH 활성이, 효소의 수준 또는 활성이 비변경된 또는 야생형 유전자의 존재 하의 수준에 비해 향상되거나 증가되도록 변경 또는 변형된 경우에, 용어 "상향조절된"이 사용된다. 특정 활성, 예를 들어 ERG9 활성이, 효소의 수준 또는 활성이 비변경된 또는 야생형 유전자의 존재 하의 수준에 비해 저하되거나 감소되도록 변경 또는 변형된 경우에, 용어 "하향조절된"이 사용된다.
용어 "과다발현하다", "과다발현하는", "과다발현된" 및 "과다발현"은 출발 미생물의 유전자 변경 전에 존재하는 것보다 더 큰 수준에서 유전자 산물의 발현, 특히 유전자 산물의 발현의 향상을 의미한다. 몇몇 실시양태에서, 미생물은 출발 미생물에 의해 생산된 것에 비해 증가된 수준에서 유전자 산물을 발현하도록 유전적으로 변경 (예를 들어, 유전자 조작)될 수 있다. 유전자 변경은 특정 유전자의 발현과 연관된 조절 서열 또는 부위의 변경 또는 변형 (예를 들어 강한 프로모터, 유도가능 프로모터 또는 다중 프로모터의 부가에 의한 또는 발현이 구성적이도록 조절 서열의 제거에 의한), 특정 유전자의 염색체 위치의 변경, 특정 유전자, 예컨대 리보솜 결합 부위 또는 전사 종결인자에 인접한 핵산 서열의 변경, 특정 유전자의 카피수의 증가, 특정 유전자의 전사 및/또는 특정 유전자 산물의 번역에 연관되는 단백질 (예를 들어, 조절 단백질, 서프레서, 인핸서, 전사 활성제 등)의 변형, 또는 관련 기술 분야에 통상적인 기술을 사용하여 특정 유전자의 발현을 탈조절하는 임의의 다른 통상적인 수단 (예를 들어, 리프레서 단백질의 발현을 차단하기 위한 안티센스 핵산 분자의 사용을 포함하고 이로 제한되지 않음)을 포함하고 이로 제한되지 않는다. 유전자 산물을 과다발현하기 위한 또 다른 방법은 그의 수명을 증가시키기 위해 유전자 산물의 안정성을 향상시키는 것이다.
용어 "탈조절된"은 미생물의 조작 전에 또는 조작되지 않은 대등한 미생물에서 발현되는 것보다 더 낮거나 더 높은 수준의 유전자 산물의 발현을 포함한다. 한 실시양태에서, 미생물은 미생물의 조작 전에 또는 조작되지 않은 대등한 미생물에서 발현되는 것보다 더 낮거나 더 높은 수준의 유전자 산물을 발현하기 위해 유전적으로 조작 (예를 들어, 유전자 조작)될 수 있다. 유전자 조작은 특정 유전자의 발현과 연관된 조절 서열 또는 부위의 변경 또는 변형 (예를 들어 강한 프로모터, 유도가능 프로모터 또는 다중 프로모터의 제거에 의한), 특정 유전자의 염색체 위치의 변경, 특정 유전자, 예컨대 리보솜 결합 부위 또는 전사 종결인자에 인접한 핵산 서열의 변경, 특정 유전자의 카피수의 증가, 특정 유전자의 전사 및/또는 특정 유전자 산물의 번역에 연관되는 단백질 (예를 들어, 조절 단백질, 서프레서, 인핸서, 전사 활성제 등)의 변형, 또는 관련 기술 분야에 통상적인 특정 유전자의 발현을 탈조절하는 임의의 다른 통상적인 수단 (예를 들어, 안티센스 핵산 분자의 사용 또는 표적 단백질의 발현을 녹-아웃 (knock-out) 또는 차단하는 다른 방법을 포함하고 이로 제한되지 않음)을 포함할 수 있고 이로 제한되지 않는다. 용어 "탈조절된 유전자 활성"은 또한 이전에 관찰되지 않은 각각의 유전자 활성, 예를 들어 라이신 데카르복실라제 활성이 바람직하게는 유전자 조작에 의해 미생물 내로 재조합 유전자, 예를 들어 이종 유전자를 하나 이상의 카피로 도입함으로써 미생물 내로 도입됨을 의미한다.
문구 "탈조절된 경로 또는 반응"은 적어도 하나의 생합성 효소의 수준 또는 활성이 변경 또는 변형되도록 생합성 경로 또는 반응의 효소를 코딩하는 적어도 하나의 유전자가 변경 또는 변형된 생합성 경로 또는 반응을 의미한다. 문구 "탈조절된 경로"는 하나 초과의 유전자가 변경 또는 변형되어, 상응하는 유전자 산물/효소의 수준 및/또는 활성을 변경하는 생합성 경로를 포함한다. 몇몇 경우에, 미생물에서 경로를 "탈조절하는" (예를 들어, 제시된 생합성 경로에서 하나 초과의 유전자를 동시에 탈조절하는) 능력은 하나 초과의 효소 (예를 들어, 2개 또는 3개의 생합성 효소)가 "클러스터" 또는 "유전자 클러스터"로 불리는 유전 물질의 인접하는 부분 상에 서로 인접하여 존재하는 유전자에 의해 코딩되는 미생물의 특정 현상에 의해 발생한다. 다른 경우에, 경로를 탈조절하기 위해, 많은 유전자가 일련의 순차적인 조작 단계에서 탈조절되어야 한다.
본 발명에 따른 탈조절된 유전자를 발현하기 위해, 폴리펩티드를 코딩하는 DNA 서열은 발현 벡터에서 전사 발현을 제어하는 조절 서열에 작동가능하게 연결된 후, 미생물 내로 도입되어야 한다. 전사 조절 서열, 예컨대 프로모터 및 인핸서에 추가로, 발현 벡터는 번역 조절 서열 및 발현 벡터를 보유하는 세포의 선택에 적합한 마커 유전자를 포함할 수 있다.
본원에서 사용되는 바와 같이, "실질적으로 순수한 단백질" 또는 화합물은 폴리아크릴아미드-나트륨 도데실 술페이트 겔 전기영동 (SDS-PAGE) 후에 단일 밴드에 의해 단백질에 대해 입증되는 바와 같이, 요구되는 정제된 단백질 또는 화합물에 본질적으로 오염시키는 세포 성분이 존재하지 않음을 의미한다. 용어 "실질적으로 순수한"은 추가로 관련 기술 분야의 통상의 기술자에 의해 사용되는 하나 이상의 순도 또는 균일성 특성에 의해 균일한 분자를 설명하고자 의도된다. 예를 들어, 실질적으로 순수한 단백질 또는 화합물은 다음과 같은 파라미터에 대한 표준 실험 편차 내에서 일정하고 재현가능한 특성을 보일 것이다: 분자량, 크로마토그래피 이동, 아미노산 조성, 아미노산 서열, 차단된 또는 비차단된 N-말단, HPLC 용리 프로파일, 생물학적 활성, 및 다른 상기 파라미터. 그러나, 상기 용어는 단백질의 인공 또는 합성 변이체 또는 2개 이상의 선택된 화합물의 인공 혼합물을 배제하는 의미가 아니다. 특히, 이 용어는 재조합 숙주로부터 단리된 융합 단백질, 예를 들어 친화도 정제를 위한 태그 (tag)에 대한 단백질의 융합체를 배제하는 의미가 아니다.
용어 2개의 핵산 서열 사이의 "서열 동일성"은 각각의 경우에 다음 파라미터를 설정한 프로그램 알고리즘 GAP (위스콘신 패키지 버전 10.0, 유니버시티 오브 위스콘신 (University of Wisconsin), 제네틱스 컴퓨터 그룹 (Genetics Computer Group) (GCG), USA 매디슨)을 사용한 정렬에 의해 계산된 전체 서열 길이에 걸친 핵산 서열의 동일성 비율을 의미하는 것으로 이해된다:
갭 가중치: 12 길이 가중치: 4
평균 매치: 2,912 평균 미스매치:-2,003
용어 2개의 아미노산 서열 사이의 "서열 동일성"은 각각의 경우에 다음 파라미터를 설정한 프로그램 알고리즘 GAP (위스콘신 패키지 버전 10.0, 유니버시티 오브 위스콘신, 제네틱스 컴퓨터 그룹 (GCG), USA 매디슨)을 사용한 정렬에 의해 계산된 전체 서열 길이에 걸친 아미노산 서열의 동일성 비율을 의미하는 것으로 이해된다:
갭 가중치: 8 길이 가중치: 2
평균 매치: 2,912 평균 미스매치:-2,003
용어 "도메인"은 진화상 관련된 단백질의 서열 정렬을 따라 특정 위치에서 보존된 아미노산의 세트를 의미한다. 다른 위치의 아미노산은 상동체 사이에서 상이할 수 있지만, 특정 위치에서 고도로 보존된 아미노산은 단백질의 구조, 안정성 또는 기능에 필수적일 가능성이 있는 아미노산을 나타낸다. 단백질 상동체 패밀리의 정렬된 서열의 높은 보존도에 의해 확인되기 때문에, 이것은 문제의 임의의 폴리펩티드가 앞서 확인된 폴리펩티드 패밀리에 속하는지 결정하기 위한 식별자로서 사용될 수 있다.
용어 "모티프" 또는 "컨센서스 서열" 또는 "시그너처"는 진화상 관련된 단백질의 서열 내의 짧은 보존된 영역을 의미한다. 모티프는 도메인의 빈번하게 고도로 보존된 부분이지만, 또한 도메인의 일부만을 포함할 수 있거나, 또는 보존된 도메인의 외부에 위치할 수 있다 (모티프의 모든 아미노산이 규정된 도메인의 외부에 위치할 경우).
도메인 확인을 위한 전문 데이타베이스, 예를 들어, SMART ([Schultz et al. (1998) Proc. Natl. Acad. Sci. USA 95, 5857-5864]; [Letunic et al. (2002) Nucleic Acids Res 30, 242-244]), InterPro (Mulder et al., (2003) Nucl. Acids. Res. 31, 315-318), Prosite ([Bucher and Bairoch (1994), A generalized profile syntax for biomolecular sequences motifs and its function in automatic sequence interpretation. (In) ISMB-94]; [Proceedings 2nd International Conference on Intelligent Systems for Molecular Biology. Altman R., Brutlag D., Karp P., Lathrop R., Searls D., Eds., pp53-61, AAAI Press, Menlo Park]; [Hulo et al., Nucl. Acids. Res. 32:D134-D137, (2004)]), 또는 Pfam ([Bateman et al., Nucleic Acids Research 30(1): 276-280 (2002)] & [The Pfam protein families database: R.D. Finn, J. Mistry, J. Tate, P. Coggill, A. Heger, J.E. Pollington, O.L. Gavin, P. Gunesekaran, G. Ceric, K. Forslund, L. Holm, E.L. Sonnhammer, S.R. Eddy, A. Bateman Nucleic Acids Research (2010) Database Issue 38:D211-222])가 존재한다. 단백질 서열의 인 실리코 분석을 위한 도구의 세트는 ExPASy 프로테오믹스 (proteomics) 서버 (스위스 인스티튜트 오브 바이오인포매틱스 (Swiss Institute of Bioinformatics) (Gasteiger et al., ExPASy: the proteomics server for in-depth protein knowledge and analysis, Nucleic Acids Res. 31:3784-3788(2003))로부터 이용가능하다. 또한, 도메인 또는 모티프는 통상적인 기술을 사용하여, 예컨대 서열 정렬에 의해 확인될 수 있다.
비교를 위한 서열의 정렬 방법은 관련 기술 분야에 잘 알려져 있고, 상기 방법은 GAP, BESTFIT, BLAST, FASTA 및 TFASTA를 포함한다. GAP는 매치의 수를 최대화하고 갭의 수를 최소화하는 2개의 서열의 전체적인 (즉, 완전 서열에 걸치는) 정렬을 발견하기 위해 문헌 [Needleman and Wunsch ((1970) J Mol Biol 48: 443-453)]의 알고리즘을 사용한다. BLAST 알고리즘 (Altschul et al. (1990) J Mol Biol 215: 403-10)은 서열 동일성 비율을 계산하고, 2개의 서열 사이의 유사성의 통계적인 분석을 수행한다. BLAST 분석을 수행하기 위한 소프트웨어는 내셔널 센터 포 바이오테크놀로지 인포메이션 (National Centre for Biotechnology Information) (NCBI)을 통해 공개적으로 이용가능하다. 상동체는 예를 들어, 디폴트 쌍별 정렬 파라미터를 사용하는 ClustalW 다중 서열 정렬 알고리즘 (버전 1.83), 및 비율 평가 방법을 사용하여 쉽게 확인될 수 있다. 또한, 전체적인 유사성 및 동일성 비율은 MatGAT 소프트웨어 패키지 (Campanella et al., BMC Bioinformatics. 2003 Jul 10;4:29. MatGAT: an application that generates similarity/identity matrices using protein or DNA sequences)에서 이용가능한 방법 중의 하나를 사용 결정될 수 있다. 작은 수동 편집은 관련 기술 분야의 통상의 기술자에게 명백할 바와 같이 보존된 모티프 사이의 정렬을 최적화하기 위해 수행될 수 있다. 또한, 상동체의 확인을 위해 전장 서열을 사용하는 대신에, 특이적 도메인이 또한 사용될 수 있다. 서열 동일성 값은 디폴트 파라미터를 사용하는 상기 언급된 프로그램을 사용하여 전체 핵산 또는 아미노산 서열에 걸쳐 또는 선택된 도메인 또는 보존된 모티프(들)에 걸쳐 결정될 수 있다. 국소 정렬을 위해, 스미스-워터맨 (Smith-Waterman) 알고리즘이 특히 유용하다 (Smith TF, Waterman MS (1981) J. Mol. Biol 147(1);195-7).
일반적으로, 이것은 임의의 서열 데이타베이스, 예컨대 공개적으로 이용가능한 NCBI 데이타베이스에 대해 질의 서열을 블라스팅 (BLASTing)하는 것을 수반하는 제1 BLAST를 포함한다. BLASTN 또는 TBLASTX (표준 디폴트 값을 이용)는 일반적으로 뉴클레오티드 서열로부터 출발할 때 사용되고, BLASTP 또는 TBLASTN (표준 디폴트 값을 이용)은 단백질 서열로부터 출발할 때 사용된다. BLAST 결과는 임의로 여과될 수 있다. 여과된 결과 또는 비-여과된 결과의 전장 서열은 이어서 질의 서열이 유래되는 유기체로부터의 서열에 대해 다시 블라스팅된다 (제2 BLAST). 이어서, 제1 및 제2 BLAST의 결과를 비교한다. 제1 BLAST로부터의 고득점 히트가 질의 서열이 유래되는 것과 동일한 종으로부터 유도되면 파라로그가 확인되고, 이상적으로는 질의 서열에서 다시 블라스팅한 한 결과가 최고 히트 중에 존재하고; 제1 BLAST의 고득점 히트가 질의 서열이 유래되는 것과 동일한 종으로부터 유도되지 않으면 오르토로그가 확인되고, 바람직하게는 질의 서열에서 다시 블라스팅한 결과가 최고 히트 중에 존재한다.
고득점 히트는 낮은 E-값을 갖는 것이다. E-값이 낮을수록, 보다 유의한 점수이다 (또는, 달리 말해서 히트가 우연히 발견될 가능성이 더 낮다). E-값의 계산은 관련 기술 분야에 잘 알려져 있다. E-값에 추가로, 비교는 또한 동일성 비율에 의해 평가된다. 동일성 비율은 특정 길이에 걸친 2개의 비교된 핵산 (또는 폴리펩티드) 서열 사이의 동일한 뉴클레오티드 (또는 아미노산)의 수를 의미한다. 큰 패밀리의 경우에, ClustalW를 사용한 후, 관련 유전자의 클러스터링의 가시화를 돕고 오르토로그 및 파라로그를 확인하기 위해 이웃 연결 계통수를 사용할 수 있다.
본원에서 규정되는 용어 "혼성화"는 실질적으로 상동성 상보성 뉴클레오티드 서열이 서로 어닐링하는 과정이다. 혼성화 과정은 전적으로 용액 내에서 발생할 수 있고, 즉, 두 상보성 핵산은 용액 내에 존재한다. 혼성화 과정은 또한 상보성 핵산 중의 하나가 매트릭스, 예컨대 자기 비드, 세파로스 비드 또는 임의의 다른 수지에 고정된 상태에서 발생할 수 있다. 혼성화 과정은 또한 상보성 핵산 중의 하나가 고체 지지체, 예컨대 니트로셀룰로스 또는 나일론 막에 고정된 또는 예를 들어 사진평판술에 의해, 예를 들어 규산질 유리 지지체에 고정된 상태에서 발생할 수 있다 (후자는 핵산 어레이 또는 마이크로어레이 또는 핵산 칩으로서 알려짐). 혼성화 발생을 허용하기 위해, 핵산 분자는 일반적으로 이중 가닥을 2개의 단일 가닥으로 용융시키기 위해 및/또는 단일 가닥 핵산으로부터 머리핀 또는 다른 2차 구조를 제거하기 위해 열에 의해 또는 화학적으로 변성된다.
용어 "엄격성"은 혼성화가 일어나는 조건을 나타낸다. 혼성화의 엄격성은 온도, 염 농도, 이온 강도 및 혼성화 완충제 조성과 같은 조건에 의해 영향받는다. 일반적으로, 저 엄격성 조건은 규정된 이온 강도 및 pH에서 특이적 서열에 대해 열 융점 (Tm)보다 약 30℃ 더 낮도록 선택된다. 중등 엄격성 조건은 온도가 Tm보다 20℃ 더 낮고, 고 엄격성 조건은 온도가 Tm보다 10℃ 더 낮다. 고 엄격성 혼성화 조건은 일반적으로 표적 핵산 서열에 높은 서열 유사성을 갖는 혼성화 서열을 단리하기 위해 사용된다. 그러나, 핵산은 서열이 차이가 날 수 있고, 유전자 코드의 축중성 때문에 실질적으로 동일한 폴리펩티드를 계속 코딩할 수 있다. 따라서, 중등 엄격성 혼성화 조건이 상기 핵산 분자를 확인하기 위해 때때로 필요할 수 있다.
Tm은 50%의 표적 서열이 완전히 매치된 프로브에 혼성화하는, 규정된 이온 강도 및 pH 하의 온도이다. Tm은 용액 상태 및 프로브의 염기 조성 및 길이에 의존한다. 예를 들어, 보다 긴 서열은 보다 높은 온도에서 특이적으로 혼성화한다. 최대 혼성화 속도는 Tm보다 약 16℃ 내지 32℃ 더 낮은 온도에서 얻는다. 혼성화 용액 내의 1가 양이온의 존재는 2개의 핵산 가닥 사이의 정전기적 척력을 감소시켜 하이브리드 형성을 촉진하고; 이러한 효과는 0.4 M 이하의 나트륨 농도에서 가시적이다 (보다 높은 농도에서, 상기 효과는 무시될 수 있다). 포름아미드는 1%의 포름아미드마다 0.6 내지 0.7℃씩 DNA-DNA 및 DNA-RNA 이중체의 융점을 저하시키고, 50% 포름아미드의 첨가는 혼성화가 30 내지 45℃에서 수행되도록 허용하지만, 혼성화 속도는 저하될 것이다. 염기쌍 미스매치는 혼성화 속도 및 이중체의 열 안정성을 감소시킨다. 평균적으로 큰 프로브에 대해, Tm은 1% 염기 미스매치당 약 1℃ 감소한다. Tm은 하이브리드의 유형에 따라 다음 식을 사용하여 계산될 수 있다:
1) DNA-DNA 하이브리드 (Meinkoth and Wahl, Anal. Biochem., 138: 267-284, 1984):
Tm= 81.5℃ + 16.6xlog10[Na+]a + 0.41x%[G/Cb] - 500x[Lc]-1 - 0.61x% 포름아미드
2) DNA-RNA 또는 RNA-RNA 하이브리드:
Tm= 79.8℃+ 18.5 (log10[Na+]a + 0.58 (%G/Cb) + 11.8 (%G/Cb)2 - 820/Lc
3) 올리고-DNA 또는 올리고-RNAd 하이브리드:
20개 미만의 뉴클레오티드에 대해: Tm= 2 (ln)
20-35개의 뉴클레오티드에 대해: Tm= 22 + 1.46 (ln)
a 또는 다른 1가 양이온에 대해, 그러나 0.01-0.4 M 범위에서만 정확함.
b 단지 30% 내지 75% 범위에서 %GC에 대해서 정확함.
c L = 염기쌍 내의 이중체의 길이.
d 올리고, 올리고뉴클레오티드; ln, = 프라이머의 효과적인 길이 = 2x(G/C의 수)+(A/T의 수).
비-특이적 결합은 많은 공지의 기술 중의 하나, 예컨대, 단백질 함유 용액을 사용한 막의 차단, 이종 RNA, DNA, 및 SDS의 혼성화 완충제에 대한 첨가, 및 Rnase를 사용한 처리를 사용하여 제어할 수 있다. 비-상동성 프로브에 대해, 일련의 혼성화가 다음 중의 하나를 변경함으로써 수행될 수 있다:
(i) 어닐링 온도의 점진적인 저하 (예를 들어 68℃에서 42℃로) 또는
(ii) 포름아미드 농도의 점진적인 저하 (예를 들어 50%로부터 0%로).
통상의 기술자는 혼성화 동안 변경될 수 있고 엄격성 조건을 유지하거나 변경할 다양한 파라미터를 잘 알고 있다. 혼성화 조건 이외에, 혼성화의 특이성은 또한 일반적으로 혼성화 후 세척의 기능에 따라 결정된다. 비특이적 혼성화에 의한 배경을 제거하기 위해, 샘플을 묽은 염 용액으로 세척한다. 상기 세척의 핵심 인자는 최종 세척 용액의 이온 강도 및 온도를 포함하고, 염 농도가 낮을수록 및 세척 온도가 높을수록, 세척 엄격성은 더 높아진다. 세척 조건은 일반적으로 혼성화 엄격성에서 또는 그 미만에서 수행된다. 양성 혼성화는 배경보다 적어도 2배의 신호를 제시한다. 일반적으로, 핵산 혼성화 검정 또는 유전자 증폭 검출 절차에 적합한 엄격한 조건은 상기 제시된 바와 같다. 더 높거나 더 낮은 엄격한 조건도 선택될 수 있다. 통상의 기술자는 세척 동안 변경될 수 있고 엄격성 조건을 유지하거나 변경할 다양한 파라미터를 알고 있다. 예를 들어, 50개 초과의 뉴클레오티드 길이의 DNA 하이브리드에 대한 전형적인 고 엄격성 혼성화 조건은 1x SSC 내의 65℃에서 또는 1x SSC 및 50% 포름아미드 내의 42℃에서의 혼성화, 이어서 0.3x SSC 내의 65℃에서의 세척을 포함한다. 50개 초과의 뉴클레오티드 길이의 DNA 하이브리드에 대한 중등 엄격성 혼성화 조건의 예는 4x SSC 내의 50℃에서 또는 6x SSC 및 50% 포름아미드 내의 40℃에서의 혼성화, 이어서 2x SSC 내의 50℃에서의 세척을 포함한다. 하이브리드의 길이는 혼성화 핵산에 대해 예상된 길이다. 서열이 알려진 핵산이 혼성화할 때, 하이브리드 길이는 서열을 정렬하고 본원에서 설명된 보존된 영역을 확인함으로써 결정될 수 있다. 1xSSC는 0.15 M NaCl 및 15 mM 시트르산나트륨이고; 혼성화 용액 및 세척 용액은 추가로 5x 덴하르트 (Denhardt) 시약, 0.5-1.0% SDS, 100 ㎍/ml의 변성된, 단편화된 연어 정자 DNA, 0.5% 피로인산나트륨을 포함할 수 있다. 엄격성 수준을 규정하기 위해, 문헌 [Sambrook et al. (2001) Molecular Cloning: a laboratory manual, 3rd Edition, Cold Spring Harbor Laboratory Press, CSH, New York] 또는 [Current Protocols in Molecular Biology, John Wiley & Sons, N.Y.] (1989 및 매년 업데이트)을 참조할 수 있다.
단백질의 "상동체"는 문제의 변형되지 않은 단백질에 비해 아미노산 치환, 결실 및/또는 삽입을 갖고 이들이 유래되는 비변경된 단백질과 유사한 생물학적 및 기능적 활성을 갖는 펩티드, 올리고펩티드, 폴리펩티드, 단백질 및 효소를 포함한다.
결실은 단백질로부터 하나 이상의 아미노산의 제거를 의미한다.
삽입은 하나 이상의 아미노산 잔기가 단백질 내의 미리 결정된 부위 내로 도입됨을 의미한다. 삽입은 N-말단 및/또는 C-말단 융합 및 단일 또는 다중 아미노산의 서열내 삽입을 포함할 수 있다. 일반적으로, 약 1 내지 10 잔기 정도의 아미노산 서열 내의 삽입은 N- 또는 C-말단 융합보다 더 작을 것이다. N- 또는 C-말단 융합 단백질 또는 펩티드의 예는 효모 2-하이브리드 시스템에서 사용되는 바와 같은 전사 활성제의 결합 도메인 또는 활성화 도메인, 파지 코트 단백질, (히스티딘)-6-태그, 글루타티온 S-트랜스퍼라제-태그, 단백질 A, 말토스-결합 단백질, 디히드로폴레이트 리덕타제, 태그·100 에피토프, c-myc 에피토프, FLAG®-에피토프, lacZ, CMP (칼모둘린-결합 펩티드), HA 에피토프, 단백질 C 에피토프 및 VSV 에피토프를 포함한다.
치환은 단백질의 아미노산을 유사한 특성 (예컨대 유사한 소수성, 친수성, 항원성, α-나선 구조 또는 β-시트 구조를 형성하거나 파괴하는 경향)을 갖는 다른 아미노산으로의 교체를 의미한다. 아미노산 치환은 일반적으로 단일 잔기 치환이지만, 폴리펩티드에 대한 기능적 제한에 따라 연속적으로 발생할 수 있고, 1 내지 10개 아미노산일 수 있고; 삽입은 대체로 약 1 내지 10개 아미노산 잔기 정도일 것이다. 아미노산 치환은 바람직하게는 보존적 아미노산 치환이다. 보존적 치환 표는 관련 기술 분야에 잘 알려져 있다 (예를 들어, 문헌 [Creighton (1984) Proteins. W.H. Freeman and Company (Eds)] 및 아래 표 1 참조).
<표 1>
본원에서 "내인성" 유전자의 언급은 그의 천연 형태로 (즉, 임의의 인간에 의한 개입이 없는) 유기체에서 발견되는 문제의 유전자를 나타낼 뿐만 아니라, 또한 후속적으로 미생물 내로 (재)도입되는 단리된 형태의 동일한 유전자 (또는 실질적으로 상동성 핵산/유전자) (트랜스진)를 나타낸다. 예를 들어, 상기 트랜스진을 함유하는 트랜스제닉 미생물은 트랜스진 발현의 실질적 감소 및/또는 내인성 유전자의 발현의 실질적 감소를 경험할 수 있다. 단리된 유전자는 유기체로부터 단리될 수 있거나 또는 예를 들어 화학적 합성에 의해 제조될 수 있다.
용어 "오르토로그" 및 "파라로그"는 유전자의 선조의 관계를 설명하기 위해 사용되는 진화 개념을 포함한다. 파라로그는 선조의 유전자의 복제를 통해 기원되는 동일한 종 내의 유전자이고; 오르토로그는 종 분화를 통해 기원되는 상이한 유기체로부터의 유전자이고 또한 공통적인 선조의 유전자로부터 유래된다.
본원에서 사용되는 용어 "스플라이스 변이체"는 선택된 인트론 및/또는 엑손이 절제, 교체, 대체 또는 부가되거나, 또는 인트론이 단축되거나 또는 연장된 핵산 서열의 변이체를 포함한다. 상기 변이체는 단백질의 생물학적 활성이 실질적으로 보유되는 것일 것이고, 이것은 단백질의 기능적 분절을 선택적으로 보유함으로써 달성될 수 있다. 상기 스플라이스 변이체는 자연에서 발견될 수 있거나 또는 제조될 수 있다. 상기 스플라이스 변이체의 예측 및 단리 방법은 관련 기술 분야에 잘 알려져 있다 (예를 들어 문헌 [Foissac and Schiex (2005) BMC Bioinformatics 6: 25] 참조).
대립유전자 또는 대립유전자 변이체는 제시된 유전자의 대체 형태이고, 동일한 염색체 위치에 존재한다. 대립유전자 변이체는 단일 뉴클레오티드 다형성 (SNP), 및 작은 삽입/결실 다형성 (INDEL)을 포함한다. INDEL의 크기는 대체로 100 bp 미만이다. SNP 및 INDEL은 대부분의 유기체의 천연 생성 다형성 균주에서 최대 서열 변이체 세트를 형성한다.
바람직한 실시양태의 상세한 설명
제1 측면에서, 본 발명은 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 및 EasG 활성의 군으로부터 선택된 적어도 하나의 활성을 포함하는 재조합 미생물을 제공하고, 여기서 EasA 활성은 EasA 리덕타제 또는 EasA 이소머라제 활성일 수 있거나 또는 EasA 리덕타제 및 EasA 이소머라제 활성일 수 있고, 재조합 미생물은 천연 맥각 알칼로이드 생산자 유기체가 아니다.
바람직하게는 재조합 미생물은 적어도 하나의 EasH 및 EasD 활성을 포함하거나, 또는 적어도 하나의 EasH 및 EasA 리덕타제 활성을 포함하거나, 또는 적어도 하나의 EasH, EasA 리덕타제 및 EasD 활성을 포함하거나, 또는 적어도 하나의 EasH 및 EasD, EasA 및 EasG 활성, 보다 더 바람직하게는 적어도 하나의 EasH, EasD, EasA 리덕타제 및 EasG 활성을 포함한다.
또 다른 실시양태에서, 재조합 미생물은 적어도 하나의 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 및 EasG 활성, 바람직하게는 적어도 하나의 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 리덕타제 및 EasG 활성을 포함한다.
추가의 실시양태에서, 재조합 미생물은 적어도 하나의 EasD, EasA 및 EasG 활성, 또는 적어도 하나의 EasD, EasA 리덕타제 및 EasG 활성, 또는 적어도 하나의 EasD, EasA 이소머라제 및 EasG 활성, 또는 적어도 하나의 EasD, EasA 리덕타제, EasA 이소머라제 및 EasG 활성을 포함한다.
재조합 미생물은 또한 적어도 하나의 DmaW, EasF, EasE, EasC, EasD, EasA 리덕타제 및 EasG 활성, 또는 하나의 DmaW, EasF, EasE, EasC, EasD, EasA 이소머라제 및 EasG 활성, 또는 적어도 하나의 DmaW, EasF, EasE, EasC, EasD, EasA 리덕타제, EasA 이소머라제 및 EasG 활성을 포함할 수 있다.
추가의 실시양태에서, 재조합 미생물은 적어도 하나의 DmaW, EasF, EasE 및 EasC 활성, 또는 적어도 하나의 DmaW, EasF, EasE, EasC 및 EasD 활성을 포함한다.
한 실시양태에서, 재조합 미생물은 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 및 EasG 활성의 군으로부터 선택된 적어도 2개의 활성을 포함하고, 여기서 EasA 활성은 EasA 리덕타제 또는 EasA 이소머라제 활성일 수 있거나 또는 EasA 리덕타제 및 EasA 이소머라제 활성일 수 있고, 여기서 상기 활성은 동일한 또는 상이한 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는 폴리펩티드에 의해 매개되고, 예를 들어, 한 실시양태에서, 재조합 미생물은 천연 맥각 알칼로이드 생산자 유기체의 하나의 종으로부터 또는 상이한 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는 적어도 EasH 및 EasD, 또는 적어도 EasH 및 EasA, 또는 적어도 EasH, EasD 및 EasA 활성을 포함한다. 추가의 예시적인 실시양태에서, 재조합 유기체는 천연 맥각 알칼로이드 생산자 유기체의 하나의 종으로부터 얻을 수 있는 적어도 EasF 및 EasE, 또는 적어도 EasF 및 EasC, 또는 적어도 EasF, EasE 및 EasC 활성을 포함한다.
천연 맥각 알칼로이드 생산자 유기체는 자연에서 발생하는 유기체이고, 적어도 하나의 유형의 맥각 알칼로이드를 생산하는 능력을 갖는다. 천연 맥각 알칼로이드 생산자 유기체는 클라비시피타세아에, 아르트로더마타세애 (Arthrodermataceae) 및 트리코코마세아에, 특히 아르트로더마 벤하미아에(Arthroderma benhamiae), 클라비셉스 종, 네오티포디움 종, 에피클로에 종, 스파셀리아 종, 발란시아 종 또는 페리글란둘라 종, 아스페르길루스 종 및 페니실리움 종, 파에실로미세스(Paecylomyces) 종, 트리코피톤(Trichophyton) 종, 아르토더마(Arthoderma) 종, 메타리지움 (Metarhizium) 종, 하이포크레아(Hypocrea) 종, 아젤로미세스(Ajellomyces) 종, 뉴로스포라(Neurospora) 종, 파라콕시디오이데스(Paracoccidioides) 종, 보트리오티니아(Botryotinia) 종에 속한다. 천연 맥각 알칼로이드 생산자 유기체의 개개의 종의 예는 본 발명의 개시내용 전체에 걸쳐 설명된다.
천연 맥각 알칼로이드 생산자 유기체 및 맥각 알칼로이드 생산을 위한 유전자 클러스터는 예를 들어 문헌 [Schardl CL, Young CA, Hesse U, Amyotte SG, Andreeva K, et al. (2013) Plant-Symbiotic Fungi as Chemical Engineers: Multi-Genome Analysis of Clavicipitaceae Reveals Dynamics of Alkaloid Loci. PLoS Genet 9(2): e1003323. doi:10.1371/journal.pgen.1003323], 및 [Schardl CL, et al.: "The epichloae: alkaloid diversity and roles in symbiosis with grasses", Curr Opin Plant Biol (2013), 16:1-9]에서 볼 수 있다.
바람직하게는, 상기 재조합 미생물은 박테리아, 효모, 방선균, 또는 사상 진균이고, 천연 맥각 알칼로이드 생산자 유기체가 아니다. 상기 사상 진균은 자낭균, 불완전균류, 또는 담자균일 수 있고, 바람직한 종은 쉬조필룸 콤뮨(Schizophyllum commune)이다.
본 발명의 숙주 세포로서 사용되는 바람직한 박테리아는 에스케리키아 콜라이(Escherichia coli) 및 바실러스 서브틸리스(Bacilus subtilis)로 이루어진 군으로부터 선택된다.
바람직한 효모는 클루이베로미세스(Kluyveromyces), 사카로미세스, 쉬조사카로미세스(Schizosaccharomyces), 야로위아(Yarrowia) 및 피키아(Pichia)로 이루어진 군으로부터 선택된다.
클루이베로미세스 락티스(Kluyveromyces lactis), 사카로미세스 세레비지아에, 쉬조사카로미세스 폼베(Schizosaccharomyces pombe), 야로위아 리폴리티카(Yarrowia lipolytica) 및 피키아 스티피테스(Pichia stipites)로 이루어진 군으로부터 선택된 재조합 미생물이 보다 바람직하다.
보다 더 바람직한 재조합 미생물은 사카로미세스 세레비지아에이다.
본 발명은 또한 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 리덕타제, EasA 이소머라제 및 EasG 활성의 군으로부터 선택된 적어도 하나의 상향조절된 활성을 갖는 재조합 천연 맥각 알칼로이드 생산자 유기체를 포함한다.
한 실시양태에서, 재조합 천연 맥각 알칼로이드 생산자 유기체는 적어도 상향조절된 EasH 및 EasD, EasA 및 EasG 활성, 보다 더 바람직하게는 상향조절된 EasH, EasD, EasA 리덕타제 및 EasG 활성을 포함한다.
또 다른 실시양태에서, 재조합 천연 맥각 알칼로이드 생산자 유기체는 적어도 상향조절된 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 및 EasG 활성, 바람직하게는 적어도 상향조절된 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 리덕타제 및 EasG 활성을 포함한다.
추가의 실시양태에서, 재조합 천연 맥각 알칼로이드 생산자 유기체는 적어도 상향조절된 EasD, EasA 및 EasG 활성, 또는 적어도 상향조절된 EasD, EasA 리덕타제 및 EasG 활성, 또는 적어도 상향조절된 EasD, EasA 이소머라제 및 EasG 활성, 또는 적어도 상향조절된 EasD, EasA 리덕타제, EasA 이소머라제 및 EasG 활성을 포함한다.
재조합 천연 맥각 알칼로이드 생산자 유기체는 또한 적어도 상향조절된 DmaW, EasF, EasE, EasC, EasD, EasA 리덕타제 및 EasG 활성, 또는 상향조절된 DmaW, EasF, EasE, EasC, EasD, EasA 이소머라제 및 EasG 활성, 또는 상향조절된 DmaW, EasF, EasE, EasC, EasD, EasA 리덕타제, EasA 이소머라제 및 EasG 활성을 포함할 수 있다.
추가의 실시양태에서, 재조합 천연 맥각 알칼로이드 생산자 유기체는 적어도 상향조절된 DmaW, EasF, EasE 및 EasC 활성, 또는 상향조절된 DmaW, EasF, EasE, EasC 및 EasD 활성을 포함한다.
또 다른 추가의 실시양태에서, 재조합 천연 맥각 알칼로이드 생산자 유기체는 EasD, EasH, EasA 리덕타제, EasA 이소머라제 및 EasG 활성의 군으로부터 선택된 적어도 하나의 하향조절된 활성을 포함한다.
한 실시양태에서, 재조합 천연 맥각 알칼로이드 생산자 유기체는 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 및 EasG 활성의 군으로부터 선택된 적어도 하나의 상향조절된 활성을 포함하고, 여기서 EasA 활성은 EasA 리덕타제 또는 EasA 이소머라제 활성일 수 있거나 또는 EasA 리덕타제 및 EasA 이소머라제 활성일 수 있고, 상기 활성은 동일한 또는 상이한 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는 폴리펩티드에 의해 매개되고, 예를 들어, 한 실시양태에서, 재조합 미생물은 천연 맥각 알칼로이드 생산자 유기체의 하나의 종으로부터 또는 상이한 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는 적어도 EasH 및 EasD, 또는 적어도 EasH 및 EasA, 또는 적어도 EasH, EasD 및 EasA 활성을 포함한다. 추가의 예시적인 실시양태에서, 재조합 유기체는 천연 맥각 알칼로이드 생산자 유기체의 하나의 종으로부터 또는 상이한 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는 적어도 EasF 및 EasE, 또는 적어도 EasF 및 EasC, 또는 적어도 EasF, EasE 및 EasC 활성을 포함한다.
바람직하게는, 재조합 천연 맥각 알칼로이드 생산자 유기체는 클라비시피타세아에, 아르트로더마타세애 및 트리코코마세아에, 특히 아르트로더마 벤하미아에, 클라비셉스 종, 네오티포디움 종, 에피클로에 종, 스파셀리아 종, 발란시아 종, 또는 페리글란둘라 종, 아스페르길루스 종, 및 페니실리움 종, 파에실로미세스 종, 트리코피톤 종, 아르트로더마 종, 메타리지움 종, 하이포크레아 종, 아젤로미세스 종, 뉴로스포라 종, 파라콕시디오이데스 종, 보트리오티니아 종의 군으로부터 선택되거나, 또는 속 아스페르길루스, 특히 아스페르길루스 자포니쿠스, 아스페르길루스 니거(Aspergillus niger), 아스페르길루스 오리자에(Aspergillus oryzae), 아스페르길루스 니둘란스(Aspergillus nidulans), 아스페르길루스 푸미가투스, 아스페르길루스 아쿨레아투스(Aspergillus aculeatus), 아스페르길루스 카에시엘루스(Aspergillus caesiellus), 아스페르길루스 칸디두스(Aspergillus candidus), 아스페르길루스 카르네우스(Aspergillus carneus), 아스페르길루스 클라바투스(Aspergillus clavatus), 아스페르길루스 데플렉투스(Aspergillus deflectus), 아스페르길루스 피셔리아누스(Aspergillus fischerianus), 아스페르길루스 플라부스(Aspergillus flavus), 아스페르길루스 글라우쿠스(Aspergillus glaucus), 아스페르길루스 오크라세우스(Aspergillus ochraceus), 아스페르길루스 파라시티쿠스(Aspergillus parasiticus), 아스페르길루스 페니실로이데스(Aspergillus penicilloides), 아스페르길루스 레스트릭투스(Aspergillus restrictus), 아스페르길루스 소자에(Aspergillus sojae), 아스페르길루스 타마리(Aspergillus tamari), 아스페르길루스 테레우스(Aspergillus terreus), 아스페르길루스 우스투스(Aspergillus ustus), 아스페르길루스 베르시콜로르(Aspergillus versicolor)로 이루어진 군으로부터 선택되고; 보다 바람직한 재조합 천연 맥각 알칼로이드 생산자 유기체는 아스페르길루스 푸미가투스, 아스페르길루스 자포니쿠스, 아스페르길루스 니둘란스 및 아스페르길루스 오리자에, 또는
속 페니실리움, 예컨대 페니실리움 아우란티오그리세움, 페니실리움 빌라이아에(Penicillium bilaiae), 페니실리움 카멤베르티(Penicillium camemberti), 페니실리움 칸디둠(Penicillium candidum), 페니실리움 크리소게눔(Penicillium chrysogenum), 페니실리움 클라비포르메(Penicillium claviforme), 페니실리움 콤뮨(Penicillium commune), 페니실리움 시트리눔(Penicillium citrinum), 페니실리움 코릴로피눔(Penicillium corylophinum), 페니실리움 코뮨(Penicillium comune), 페니실리움 펠루타눔(Penicillium fellutanum), 페니실리움 와크스마니이(Penicillium waksmanii), 페니실리움 크루스토숨(Penicillium crustosum), 페니실리움 디기타툼(Penicillium digitatum), 페니실리움 엑스판숨(Penicillium expansum), 페니실리움 푸니쿨로숨(Penicillium funiculosum), 페니실리움 글라브룸(Penicillium glabrum), 페니실리움 글라우쿰, 페니실리움 이탈리쿰(Penicillium italicum), 페니실리움 라쿠싸르미엔테이(Penicillium lacussarmientei), 페니실리움 마르네페이(Penicillium marneffei), 페니실리움 푸르푸로게눔(Penicillium purpurogenum), 페니실리움 로쿠에포르티(Penicillium roqueforti), 페니실리움 스톨로니페룸(Penicillium stoloniferum), 페니실리움 울라이엔세(Penicillium ulaiense), 페니실리움 베루코숨(Penicillium verrucosum), 페니실리움 비리디카툼(Penicillium viridicatum); 또는
속 클라비셉스, 예컨대 클라비셉스 아프리카나(Claviceps africana), 클라비셉스 기간테아(Claviceps gigantea), 클라비셉스 그로히이(Claviceps grohii), 클라비셉스 사이페리(Claviceps cyperi), 클라비셉스 푸시포르미스(Claviceps fusiformis), 클라비셉스 히르텔라(Claviceps hirtella), 클라비셉스 니그리칸스(Claviceps nigricans), 클라비셉스 파스팔리(Claviceps paspali), 클라비셉스 푸르푸레아, 클라비셉스클라비셉스 소르기(Claviceps sorghi), 클라비셉스 지자니아에(Claviceps zizaniae), 바람직하게는 클라비셉스 아프리카나, 클라비셉스 파스팔리, 또는 클라비셉스 푸르푸레아, 또는
속 파에실로미세스, 예컨대 파에실로미세스 디바리카투스, 속 아르트로더마, 예컨대 아르트로더마 벤하미아에, 또는 속 트리코피톤, 예컨대 트리코피톤 베루코숨(Trichophyton verrucosum), 또는 속 미크로스포룸(Microsporum), 예컨대 미크로스포룸 카니스(Microsporum canis)
로 이루어진 군으로부터 선택된다.
아스페르길루스 자포니쿠스, 아스페르길루스 니거, 아스페르길루스 오리자에, 아스페르길루스 니둘란스, 아스페르길루스 푸미가투스, 클라비셉스 아프리카나, 클라비셉스 푸르푸레아, 클라비셉스 푸시포르미스, 클라비셉스 파스팔리, 클라비셉스 지자니에아(Claviceps zizaniea), 페니실리움 크리소게눔, 페니실리움 시트리눔, 페니실리움 코릴로피눔, 페니실리움 펠루타눔, 페니실리움 와크스마니이, 페니실리움 로쿠에포르티, 및 파에실로미세스 디바리카투스로 이루어진 군으로부터 선택되는, 바람직하게는 바이러스 아스페르길루스 자포니쿠스, 아스페르길루스 푸미가투스, 클라비셉스 푸르푸레아, 클라비셉스 파스팔리, 클라비셉스 아프리카나, 및 파에실로미세스 디바리카투스로 이루어진 군으로부터 선택되는 재조합 천연 맥각 알칼로이드 생산자 유기체가 보다 더 바람직하다.
재조합 천연 맥각 알칼로이드 생산자 유기체는 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드, 또는 카노클라빈 I 중 적어도 하나를 생산하기 위해 사용될 수 있고, 보다 많은 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드, 및/또는 카노클라빈 I을 제공하기 위해 활성이 상향조절 및/또는 하향조절되어야 하는지에 대한 추가의 지침은 도 1 및 도 2에 포함되어 있다. 예를 들어, 재조합 천연 맥각 알칼로이드 생산자 유기체는 상향조절된 EasH 활성을 갖는, 바람직하게는 상향조절된 EasH 및 EasA 리덕타제 활성 및 하향조절된 EasA 이소머라제 활성을 갖는 클라비셉스 파스팔리, 클라비셉스 푸르푸레아, 또는 클라비셉스 아리카나(Claviceps aricana) 세포이다.
상기 설명한 모든 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체는 바람직하게는 트립토판 및/또는 DMAPP의 확대된 세포 내부 공급원 및/또는 Me-DMAT의 확대된 세포 내부 공급원을 포함한다. 상기 DMAPP 및/또는 Me-DMAT의 확대된 세포 내부 공급원은 메발로네이트 경로의 유전자 또는 폴리펩티드 또는 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체의 활성을 상향조절함으로써 제공될 수 있다. 사카로미세스 세레비지아에의 메발로네이트 경로의 유전자 및 폴리펩티드의 예는 서열 12, 13, 14, 15, 16, 17, 또는 18에 의해 설명된다. 상기 유전자의 상동체 및 그의 서열 변이체는 관련 기술 분야의 통상의 기술자에게 쉽게 이용가능하다.
예를 들어, 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체는 ERG9 및 ERG20 활성의 군으로부터 선택된 적어도 하나의 하향조절된 활성을 포함할 수 있거나, 또는 적어도 하나의 상향조절된 HMG-CoA 리덕타제 활성을 포함할 수 있거나, 또는 ERG9 및 ERG20 활성의 군으로부터 선택된 적어도 하나의 하향조절된 활성 및 적어도 하나의 상향조절된 HMG-CoA 리덕타제 활성을 포함할 수 있다.
따라서, 본 발명은 바람직하게는, 본 발명의 폴리뉴클레오티드에 추가로, 본원에서 언급되는 클라빈-유형 알칼로이드의 합성을 위한 출발 물질로서의 트립토판의 합성을 위해 필요하거나 이를 용이하게 하는 폴리뉴클레오티드를 포함하는 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체를 고려한다. 보다 바람직하게는, 그러한 효소는 트립토판신타제이다. 트립토판의 생합성에 대한 보다 상세한 내용은 예를 들어 문헌 [Radwanski 1995, Plant Cell 7(7): 921-934]에서 볼 수 있다.
본 발명의 한 실시양태에서, 상기 설명한 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체는 적어도 하나의 EasH 활성을 코딩하는, 또는 적어도 하나의 EasH 및 EasD 활성을 코딩하는, 또는 적어도 하나의 EasA 리덕타제 또는 적어도 하나의 EasA 이소머라제 활성을 코딩하는, 또는 적어도 하나의 EasD, EasA 이소머라제 및 EasG 활성을 코딩하는, 또는 적어도 하나의 EasD, EasA 리덕타제 및 EasG 활성을 코딩하는, 또는 적어도 하나의 EasH, EasD, EasA 리덕타제, EasA 이소머라제 및 EasG 활성을 코딩하는, 또는 적어도 하나의 EasH, EasD, EasA 리덕타제 및 EasG 활성을 코딩하는, 또는 적어도 하나의 EasH, EasD, EasA 이소머라제 및 EasG 활성을 코딩하는, 또는 적어도 하나의 DmaW, EasF, EasE 및 EasC 활성을 코딩하는, 또는 상기 설명한 것 중 적어도 2개를 코딩하는 적어도 하나의 재조합 폴리뉴클레오티드를 포함한다.
따라서, 본 발명의 추가의 실시양태는 적어도 하나의 EasH 활성을 코딩하는, 또는 적어도 하나의 EasH 및 EasD 활성을 코딩하는, 또는 적어도 하나의 EasA 리덕타제 또는 적어도 하나의 EasA 이소머라제 활성을 코딩하는, 또는 적어도 하나의 EasD, EasA 이소머라제 및 EasG 활성을 코딩하는, 또는 적어도 하나의 EasD, EasA 리덕타제 및 EasG 활성을 코딩하는, 또는 적어도 하나의 EasH, EasD, EasA 리덕타제, EasA 이소머라제 및 EasG 활성을 코딩하는, 또는 적어도 하나의 EasH, EasD, EasA 리덕타제 및 EasG 활성을 코딩하는, 또는 적어도 하나의 EasH, EasD, EasA 이소머라제 및 EasG 활성을 코딩하는, 또는 적어도 하나의 DmaW, EasF, EasE 및 EasC 활성을 코딩하는, 또는 상기 설명한 것 중 적어도 2개를 코딩하는 재조합 폴리뉴클레오티드이다.
본 발명을 위해 설명된 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드 중의 임의의 하나의 DmaW 활성은 바람직하게는 다음 군으로부터 선택된 적어도 하나의 폴리펩티드 또는 적어도 하나의 폴리펩티드를 코딩하는 적어도 하나의 폴리뉴클레오티드에 의한 것이다:
a) 서열 145 및 146에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
b) 서열 147 및 또는 190에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
c) 서열 1, 19, 20 179, 및/또는 180에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
d) 서열 1, 19, 20, 21, 22, 23, 24, 25, 26, 27 및/또는 28에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
e) 서열 102, 122 및/또는 123에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드,
f) 서열 129 및/또는 137에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드,
g) 서열 102, 122, 123, 129 및/또는 137에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드,
h) 엄격한 조건 하에 서열 102, 122, 123, 129 및/또는 137에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드,
i) a) 내지 h) 중 적어도 2개, 바람직하게는 적어도 b) 및 c)에 따른 폴리펩티드 및
j) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 i) 중 적어도 하나에 따른 폴리펩티드.
DmaW 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드는 원칙적으로, 본원에 언급된 임의의 천연 맥각 알칼로이드 생산자로부터 얻을 수 있다. 한 실시양태에서, 폴리뉴클레오티드는
아스페르길루스 테레우스, 발란시아 안드로포고니스(Balansia andropogonis), 발란시아 사이페리(Balansia cyperi )), 발란시아 오브텍타(Balansia obtecta), 발란시아 필루아에포르미스(Balansia pilulaeformis), 클라비셉스 아프리카나, 클라비셉스 푸시포르미스, 클라비셉스 히르텔라, 스파셀리아 에리오클로아에(Sphacelia eriochloae), 스파셀리아 텍센시스(Sphacelia texensis), 스파셀리아 로벨레시이(Sphacelia lovelessii), 네오티포디움 종 Lp1, 네오티포디움 코에노피알룸(Neotyphodium coenophialum), 네오티포디움 롤리이, 페니실리움 시트리눔, 페니실리움 콤뮨, 페니실리움 코릴로필룸(Penicillium corylophillum), 페니실리움 크루스토숨, 페니실리움 펠루타눔 및 페니실리움 팔리탄스(Penicillium palitans)로 이루어지는 군, 또는
아스페르길루스 자포니쿠스, 아스페르길루스 니거, 아스페르길루스 오리자에, 아스페르길루스 니둘란스, 아스페르길루스 푸미가투스, 클라비셉스 푸르푸레아, 클라비셉스 푸시포르미스, 클라비셉스 파스팔리, 클라비셉스 지자니아에, 페니실리움 크리소게눔, 페니실리움 시트리눔, 페니실리움 코릴로피눔, 페니실리움 펠루타눔, 페니실리움 와크스마니이, 페니실리움 로쿠에포르티 및 파에실로미세스 디바리카투스로 이루어지는 군, 또는
아르트로더마 벤타미아에(Arthroderma benthamiae), 아르트로더마 오타에(Arthroderma otae), 에피클로에 브라키엘리트리(Epichloe brachyelytri), 에피클로에 페스투카에(Epichloe festucae), 에피클로에 글리세리아에(Epichloe glyceriae), 에피클로에 티프니아(Epichloe typhina), 말브란케아 아우란티아카(Malbranchea aurantiaca), 미크로스포룸 카니스, 메타리지움 아크리둠(Metarhizium acridum), 메타리지움 로베르트시이(Metarhizium robertsii), 페리글란둘라 이포모에아에(Periglandula ipomoeae), 트리코피톤 에퀴늄(Trichophyton equinum), 및 트리코피톤 톤수란스(Trichophyton tonsurans)로 이루어지는 군, 또는
아스페르길루스 자포니쿠스, 아스페르길루스 푸미가투스, 클라비셉스 푸르푸레아, 및 파에실로미세스 디바리카투스로 이루어지는 군, 또는
상기 군의 임의의 하나의 조합인 군으로 이루어지는 군
으로부터 선택되는 천연 맥각 알칼로이드 생산자로부터 얻어진다.
DmaW 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드는 본원에 개시된 DmaW 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드에 대한 서열 정보에 의해 제공되는 서열 정보를 기촐로 하여 설계된 PCR-프라이머를 사용하는 PCR-방법을 통해 게놈 단편 또는 cDNA 단편을 증폭함으로써 상기 나열된 유기체로부터 얻을 수 있다. 대체로, 상기 방법에 사용되는 PCR-프라이머의 세트는 DmaW 활성을 갖고 본원에 개시된 폴리펩티드를 코딩하는 서열의 적어도 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 28개, 그러나 100, 90, 80, 70, 60, 50, 40 또는 30개 미만의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머로 이루어진다. 바람직하게는, PCR-프라이머는 증폭할 폴리뉴클레오티드의 서열에 대한 임의의 뉴클레오티드 미스매치가 없이 결합할 것이다. 동일한 방법은 또한 각각의 활성을 갖는 폴리펩티드에 대해 본원에서 제공되는 서열 정보를 사용하여 EasF, EasE, EasC, EasD, EasH, EasA 및 EasG 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드의 증폭에 적용될 것이다.
바람직하게는, 적어도 하나의 DmaW 활성을 갖는 폴리펩티드는 표 2에 나열된 서열, 또는 상기 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리펩티드로부터 선택된다.
<표 2>
바람직하게는, DmaW 활성을 갖는 폴리펩티드는 아래에서 설명되는 적어도 하나의 DmaW 시그너처 서열을 포함하거나 또는 DmaW 컨센서스 서열을 포함한다. 바람직하게는, 이것은 두 DmaW 시그너처 서열을 모두 포함하고, 심지어 두 DmaW 시그너처 서열 및 DmaW 컨센서스 서열을 포함한다. DmaW 시그너처 및 컨센서스 서열은 각각의 개별적인 아미노산이 관련 기술 분야에서 사용되는 단일 문자 아미노산 코드에 의해 특정되는 연속적인 아미노산 서열을 설명한다. "X"로 특정된 아미노산은 임의의 아미노산을 나타내고, 바람직하게는 표 2에 표시되고 도 3a 내지 3d에 제시된 서열의 DmaW 서열 정렬의 적어도 하나의 아미노산 서열에서 동일한 위치를 갖는 아미노산을 나타낸다.
DmaW 시그너처 서열 1 (서열 124):
DmaW 시그너처 서열 2 (서열 125):
DmaW 컨센서스 서열 (서열 126):
DmaW 컨센서스 서열 2 (서열 190):
본 발명을 위해 설명된 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드의 DmaW 활성은 다음 군으로부터 선택된 적어도 하나의 폴리펩티드 또는 적어도 하나의 폴리펩티드를 코딩하는 적어도 하나의 폴리뉴클레오티드의 활성에 의한 것이다:
a) 서열 19에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
b) 서열 19에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 190에 적어도 85%, 바람직하게는 적어도 89% 서열 동일성을 갖고, 보다 더 바람직하게는 도 3에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는 폴리펩티드,
c) 서열 180에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
d) 서열 180에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 190에 적어도 85%, 바람직하게는 적어도 89% 서열 동일성을 갖고, 보다 더 바람직하게는 도 3에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는 폴리펩티드, 및
e) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 d) 중 적어도 하나에 따른 폴리펩티드.
본 발명을 위해 설명된 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드 중의 임의의 하나의 EasF 활성은 바람직하게는 다음 군으로부터 선택된 적어도 하나의 폴리펩티드 또는 적어도 하나의 폴리펩티드를 코딩하는 적어도 하나의 폴리뉴클레오티드의 활성에 의한 것이다:
a) 서열 154에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
b) 서열 6, 75 및/또는 76에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
c) 서열 6, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84 및/또는 85에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
d) 서열 107 및/또는 120에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드,
e) 서열 134 및/또는 142에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드,
f) 서열 107, 120, 134 및/또는 142에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
g) 엄격한 조건 하에 서열 107, 120, 134 및/또는 142에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드,
h) a) 내지 g) 중 적어도 2개, 바람직하게는 적어도 a) 및 b)에 따른 폴리펩티드 및
i) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 h) 중 적어도 하나에 따른 폴리펩티드.
EasF 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드는 원칙적으로, 본원에 언급된 임의의 천연 맥각 알칼로이드 생산자로부터 얻을 수 있다. 한 실시양태에서, 폴리뉴클레오티드는
아스페르길루스 테레우스, 발란시아 안드로포고니스, 발란시아 사이페리, 발란시아 오브텍타, 발란시아 필루아에포르미스, 클라비셉스 아프리카나, 클라비셉스 푸시포르미스, 클라비셉스 히르텔라, 스파셀리아 에리오클로아에, 스파셀리아 텍센시스, 스파셀리아 로벨레시이, 네오티포디움 종 Lp1, 네오티포디움 코에노피알룸, 네오티포디움 롤리이, 페니실리움 시트리눔, 페니실리움 콤뮨, 페니실리움 코릴로필룸, 페니실리움 크루스토숨, 페니실리움 펠루타눔 및 페니실리움 팔리탄스로 이루어지는 군, 또는
아스페르길루스 자포니쿠스, 아스페르길루스 니거, 아스페르길루스 오리자에, 아스페르길루스 니둘란스, 아스페르길루스 푸미가투스, 클라비셉스 푸르푸레아, 클라비셉스 푸시포르미스, 클라비셉스 파스팔리, 클라비셉스 지자니아에, 페니실리움 크리소게눔, 페니실리움 시트리눔, 페니실리움 코릴로피눔, 페니실리움 펠루타눔, 페니실리움 와크스마니이, 페니실리움 로쿠에포르티 및 파에실로미세스 디바리카투스로 이루어지는 군, 또는
아르트로더마 벤타미아에, 아르트로더마 깁세움(Arthroderma gypseum), 아르트로더마 오타에, 에피클로에 브라키엘리트리, 에피클로에 페스투카에, 에피클로에 글리세리아에, 에피클로에 티프니아, 말브란케아 아우란티아카, 미크로스포룸 카니스, 메타리지움 아크리둠, 메타리지움 로베르트시이, 페리글란둘라 이포모에아에, 트리코피톤 에퀴늄, 트리코피톤 루브룸(Trichophyton rubrum), 트리코피톤 톤수란스, 및 트리코피톤 베루코숨으로 이루어지는 군, 또는
아스페르길루스 자포니쿠스, 아스페르길루스 푸미가투스, 클라비셉스 푸르푸레아, 및 파에실로미세스 디바리카투스로 이루어지는 군, 또는
상기 군의 임의의 하나의 조합인 군으로 이루어지는 군
으로부터 선택된 천연 맥각 알칼로이드 생산자로부터 얻어진다.
바람직하게는, EasF 활성을 갖는 적어도 하나의 폴리펩티드는 표 3에 나열된 서열, 또는 상기 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리펩티드로부터 선택된다.
<표 3>
바람직하게는, EasF 활성을 갖는 폴리펩티드는 EasF 컨센서스 서열을 포함한다. EasF 컨센서스 서열은 각각의 개별적인 아미노산이 관련 기술 분야에서 사용되는 단일 문자 아미노산 코드에 따라 특정되는 연속적인 아미노산 서열을 설명한다. "X"로 특정된 아미노산은 임의의 아미노산을 나타내고, 바람직하게는 표 3에 표시되고 도 4a 내지 4c에 제시된 서열의 EasF 서열 정렬의 적어도 하나의 아미노산 서열에서 동일한 위치를 갖는 아미노산을 나타낸다.
EasF 시그너처 서열 (서열 154):
바람직하게는, 본 발명을 위해 설명된 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드의 EasF 활성은 다음 군으로부터 선택된 적어도 하나의 폴리펩티드 또는 적어도 하나의 폴리펩티드를 코딩하는 적어도 하나의 폴리뉴클레오티드의 활성에 의한 것이다:
a) 서열 6에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
b) 서열 6에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 154에 적어도 75%, 바람직하게는 적어도 79% 서열 동일성을 갖고, 보다 더 바람직하게는 도 4에 제시된 정렬에 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는 폴리펩티드,
c) 서열 75에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
d) 서열 6에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 154에 적어도 75%, 바람직하게는 적어도 83% 서열 동일성을 갖고, 보다 더 바람직하게는 도 4에 제시된 정렬에 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는 폴리펩티드,
e) 서열 76에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
f) 서열 76에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 154에 적어도 75%, 바람직하게는 적어도 78% 서열 동일성을 갖고, 보다 더 바람직하게는 도 4에 제시된 정렬에 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는 폴리펩티드, 및
g) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 f) 중 적어도 하나에 따른 폴리펩티드.
본 발명을 위해 설명된 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드 중의 임의의 하나의 EasE 활성은 바람직하게는 다음 군으로부터 선택된 적어도 하나의 폴리펩티드 또는 적어도 하나의 폴리뉴클레오티드의 활성에 의한 것이다:
a) 서열 153, 185 및/또는 191에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는, 바람직하게는 서열 191에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
b) 서열 5 및/또는 64에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
c) 서열 5, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73 및/또는 74에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 또는 서열 5, 64, 175, 66, 67, 68, 69, 176, 71, 177, 73 및/또는 178에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
d) 서열 106에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드,
e) 서열 133 및/또는 141에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드,
f) 서열 106, 133 및/또는 141에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드,
g) 엄격한 조건 하에 서열 106, 133 및/또는 141에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드,
h) a) 내지 g) 중 적어도 2개, 바람직하게는 적어도 a) 및 b)에 따른 폴리펩티드 및
i) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 h) 중 적어도 하나에 따른 폴리펩티드.
바람직하게는, 코딩된 폴리펩티드는 소포체로의 분류 신호로 기능할 수 있는 N-말단 서열을 갖는다.
EasE 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드는 원칙적으로 본원에 언급된 임의의 천연 맥각 알칼로이드 생산자로부터 얻을 수 있다. 한 실시양태에서, 폴리뉴클레오티드는
아스페르길루스 테레우스, 발란시아 안드로포고니스, 발란시아 사이페리, 발란시아 오브텍타, 발란시아 필루아에포르미스, 클라비셉스 아프리카나, 클라비셉스 푸시포르미스, 클라비셉스 히르텔라, 스파셀리아 에리오클로아에, 스파셀리아 텍센시스, 스파셀리아 로벨레시이, 네오티포디움 종 Lp1, 네오티포디움 코에노피알룸, 네오티포디움 롤리이, 페니실리움 시트리눔, 페니실리움 콤뮨, 페니실리움 코릴로필룸, 페니실리움 크루스토숨, 페니실리움 펠루타눔 및 페니실리움 팔리탄스로 이루어지는 군, 또는
아스페르길루스 자포니쿠스, 아스페르길루스 니거, 아스페르길루스 오리자에, 아스페르길루스 니둘란스, 아스페르길루스 푸미가투스, 클라비셉스 푸르푸레아, 클라비셉스 푸시포르미스, 클라비셉스 파스팔리, 클라비셉스 지자니아에, 페니실리움 크리소게눔, 페니실리움 시트리눔, 페니실리움 코릴로피눔, 페니실리움 펠루타눔, 페니실리움 와크스마니이, 페니실리움 로쿠에포르티 및 파에실로미세스 디바리카투스로 이루어지는 군, 또는
아르트로더마 벤타미아에, 아르트로더마 깁세움, 아르트로더마 오타에, 에피클로에 브라키엘리트리, 에피클로에 페스투카에, 에피클로에 글리세리아에, 에피클로에 티프니아, 말브란케아 아우란티아카, 미크로스포룸 카니스, 메타리지움 아크리둠, 메타리지움 로베르트시이, 페리글란둘라 이포모에아에, 트리코피톤 에퀴늄, 트리코피톤 루브룸, 트리코피톤 톤수란스, 및 트리코피톤 베루코숨으로 이루어지는 군, 또는
아스페르길루스 자포니쿠스, 아스페르길루스 푸미가투스, 클라비셉스 푸르푸레아, 및 파에실로미세스 디바리카투스로 이루어지는 군, 또는
상기 군의 임의의 하나의 조합인 군으로 이루어지는 군
으로부터 선택된 천연 맥각 알칼로이드 생산자로부터 얻어진다.
바람직하게는, EasE 활성을 갖는 적어도 하나의 폴리펩티드는 표 4에 나열된 서열, 또는 상기 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리펩티드로부터 선택된다.
<표 4>
바람직하게는, EasE 활성을 갖는 폴리펩티드는 EasE 컨센서스 서열을 포함한다. EasE 컨센서스 서열은 각각의 개별적인 아미노산이 관련 기술 분야에서 사용되는 단일 문자 아미노산 코드에 따라 특정되는 연속적인 아미노산 서열을 설명한다. "X"로 특정된 아미노산은 임의의 아미노산을 나타내고, 바람직하게는 표 4에 표시되고 도 5a 내지 5e에 제시된 서열의 EasE 서열 정렬의 적어도 하나의 아미노산 서열에서 동일한 위치를 갖는 아미노산을 나타낸다.
EasE 시그너처 서열 (서열 153):
EasE 컨센서스 서열 2 (서열 153):
EasE 코어 컨센서스 서열 (서열 191):
바람직하게는, 본 발명을 위해 설명된 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드의 EasE 활성은 다음 군으로부터 선택된 적어도 하나의 폴리펩티드 또는 적어도 하나의 폴리펩티드를 코딩하는 적어도 하나의 폴리뉴클레오티드의 활성에 의한 것이다:
a) 서열 5에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
b) 서열 5에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 191에 적어도 70%, 바람직하게는 적어도 73% 서열 동일성을 갖고, 보다 더 바람직하게는 도 5에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는 폴리펩티드,
c) 서열 64에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
d) 서열 64에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 191에 적어도 75%, 바람직하게는 적어도 79% 서열 동일성을 갖고, 보다 더 바람직하게는 도 5에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는 폴리펩티드, 및
e) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 d) 중 적어도 하나에 따른 폴리펩티드.
본 발명을 위해 설명된 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드 중의 임의의 하나의 EasC 활성은 바람직하게는 다음 군으로부터 선택된 적어도 하나의 폴리펩티드 또는 적어도 하나의 폴리뉴클레오티드의 활성에 의한 것이다:
a) 서열 151 및/또는 192에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는,
b) 서열 3 및/또는 41에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
c) 서열 3 또는 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51 및/또는 52에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 104에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
e) 서열 131 또는 139에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는,
f) 서열 104, 131 및/또는 139에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
g) 엄격한 조건 하에 서열 104, 131 및/또는 139에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드 또는
h) a) 내지 g) 중 적어도 2개, 바람직하게는 적어도 a) 및 b)에 따른 폴리펩티드 및
i) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 h) 중 적어도 하나에 따른 폴리펩티드.
EasC 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드는 원칙적으로 본원에 언급된 임의의 천연 맥각 알칼로이드 생산자로부터 얻을 수 있다. 한 실시양태에서, 폴리뉴클레오티드는
아스페르길루스 테레우스, 발란시아 안드로포고니스, 발란시아 사이페리, 발란시아 오브텍타, 발란시아 필루아에포르미스, 클라비셉스 아프리카나, 클라비셉스 푸시포르미스, 클라비셉스 히르텔라, 스파셀리아 에리오클로아에, 스파셀리아 텍센시스, 스파셀리아 로벨레시이, 네오티포디움 종 Lp1, 네오티포디움 코에노피알룸, 네오티포디움 롤리이, 페니실리움 시트리눔, 페니실리움 콤뮨, 페니실리움 코릴로필룸, 페니실리움 크루스토숨, 페니실리움 펠루타눔 및 페니실리움 팔리탄스로 이루어지는 군, 또는
아스페르길루스 자포니쿠스, 아스페르길루스 니거, 아스페르길루스 오리자에, 아스페르길루스 니둘란스, 아스페르길루스 푸미가투스, 클라비셉스 푸르푸레아, 클라비셉스 푸시포르미스, 클라비셉스 파스팔리, 클라비셉스 지자니아에, 페니실리움 크리소게눔, 페니실리움 시트리눔, 페니실리움 코릴로피눔, 페니실리움 펠루타눔, 페니실리움 와크스마니이, 페니실리움 로쿠에포르티 및 파에실로미세스 디바리카투스로 이루어지는 군, 또는
아르트로더마 벤타미아에, 아르트로더마 깁세움, 아르트로더마 오타에, 에피클로에 브라키엘리트리, 에피클로에 페스투카에, 에피클로에 글리세리아에, 에피클로에 티프니아, 메타리지움 아크리둠, 메타리지움 로베르트시이, 페리글란둘라 이포모에아에, 트리코피톤 에퀴늄, 트리코피톤 루브룸 및 트리코피톤 베루코숨으로 이루어지는 군, 또는
아스페르길루스 자포니쿠스, 아스페르길루스 푸미가투스, 클라비셉스 푸르푸레아, 및 파에실로미세스 디바리카투스로 이루어지는 군, 또는
상기 군의 임의의 하나의 조합인 군으로 이루어지는 군
으로부터 선택된 천연 맥각 알칼로이드 생산자로부터 얻어진다.
바람직하게는, EasC 활성을 갖는 적어도 하나의 폴리펩티드는 표 5에 나열된 서열, 또는 상기 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리펩티드로부터 선택된다.
<표 5>
바람직하게는, EasC 활성을 갖는 폴리펩티드는 EasC 컨센서스 서열을 포함한다. EasC 컨센서스 서열은 각각의 개별적인 아미노산이 관련 기술 분야에서 사용되는 단일 문자 아미노산 코드에 따라 특정되는 연속적인 아미노산 서열을 설명한다. "X"로 특정된 아미노산은 임의의 아미노산을 나타내고, 바람직하게는 표 5에 표시되고 도 6a 내지 6d에 제시된 서열의 EasC 서열 정렬의 적어도 하나의 아미노산 서열에서 동일한 위치를 갖는 아미노산을 나타낸다.
EasC 컨센서스 서열 (서열 151):
EasC 컨센서스 코어 서열 (서열 192):
바람직하게는, 본 발명을 위해 설명된 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드의 EasC 활성은 다음 군으로부터 선택된 적어도 하나의 폴리펩티드 또는 적어도 하나의 폴리펩티드를 코딩하는 적어도 하나의 폴리뉴클레오티드의 활성에 의한 것이다:
a) 서열 3에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
b) 서열 3에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 192에 적어도 85%, 바람직하게는 적어도 87% 서열 동일성을 갖고, 보다 더 바람직하게는 도 6에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는 폴리펩티드,
c) 서열 41에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
d) 서열 41에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 192에 적어도 85%, 바람직하게는 적어도 90% 서열 동일성을 갖고, 보다 더 바람직하게는 도 6에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는 폴리펩티드, 및
e) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 d) 중 적어도 하나에 따른 폴리펩티드.
본 발명을 위해 설명된 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드 중의 임의의 하나의 EasD 활성은 바람직하게는 다음 군으로부터 선택된 적어도 하나의 폴리펩티드 또는 적어도 하나의 폴리뉴클레오티드의 활성에 의한 것이다:
a) 서열 152에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는,
b) 서열 4 및/또는 53에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
c) 서열 4, 53, 54, 55, 56, 57, 58, 59, 60, 62 및/또는 63에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 105에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
e) 서열 132 및/또는 140에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
f) 서열 105, 132 및/또는 140에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드,
g) 엄격한 조건 하에 서열 105, 132 및/또는 140에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
h) a) 내지 g) 중 적어도 2개, 바람직하게는 적어도 a) 및 b)에 따른 폴리펩티드 및
i) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 h) 중 적어도 하나에 따른 폴리펩티드.
EasD 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드는 원칙적으로 본원에 언급된 임의의 천연 맥각 알칼로이드 생산자로부터 얻을 수 있다. 한 실시양태에서, 폴리뉴클레오티드는
아스페르길루스 테레우스, 발란시아 안드로포고니스, 발란시아 사이페리, 발란시아 오브텍타, 발란시아 필루아에포르미스, 클라비셉스 아프리카나, 클라비셉스 푸시포르미스, 클라비셉스 히르텔라, 스파셀리아 에리오클로아에, 스파셀리아 텍센시스, 스파셀리아 로벨레시이, 네오티포디움 종 Lp1, 네오티포디움 코에노피알룸, 네오티포디움 롤리이, 페니실리움 시트리눔, 페니실리움 콤뮨, 페니실리움 코릴로필룸, 페니실리움 크루스토숨, 페니실리움 펠루타눔 및 페니실리움 팔리탄스로 이루어지는 군, 또는
아스페르길루스 자포니쿠스, 아스페르길루스 니거, 아스페르길루스 오리자에, 아스페르길루스 니둘란스, 아스페르길루스 푸미가투스, 클라비셉스 푸르푸레아, 클라비셉스 푸시포르미스, 클라비셉스 파스팔리, 클라비셉스 지자니아에, 페니실리움 크리소게눔, 페니실리움 코릴로피눔, 페니실리움 펠루타눔, 페니실리움 와크스마니이, 페니실리움 로쿠에포르티 및 파에실로미세스 디바리카투스로 이루어지는 군, 또는
아르트로더마 벤타미아에, 아르트로더마 깁세움, 아르트로더마 오타에, 에피클로에 브라키엘리트리, 에피클로에 페스투카에, 에피클로에 글리세리아에, 에피클로에 티프니아, 미크로스포룸 카니스, 메타리지움 아크리둠, 메타리지움 로베르트시이, 페리글란둘라 이포모에아에, 트리코피톤 에퀴늄, 트리코피톤 루브룸, 트리코피톤 톤수란스, 트리코피톤 베루코숨 및 트리코더마 비렌스 (Trichoderma virens)로 이루어지는 군, 또는
아스페르길루스 자포니쿠스, 아스페르길루스 푸미가투스, 클라비셉스 푸르푸레아, 및 파에실로미세스 디바리카투스로 이루어지는 군, 또는
상기 군의 임의의 하나의 조합인 군으로 이루어지는 군
으로부터 선택된 천연 맥각 알칼로이드 생산자로부터 얻어진다.
바람직하게는, EasD 활성을 갖는 적어도 하나의 폴리펩티드는 표 6에 나열된 서열, 또는 상기 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리펩티드로부터 선택된다.
<표 6>
바람직하게는, EasD 활성을 갖는 폴리펩티드는 EasD 컨센서스 서열을 포함한다. EasD 컨센서스 서열은 각각의 개별적인 아미노산이 관련 기술 분야에서 사용되는 단일 문자 아미노산 코드에 따라 특정되는 연속적인 아미노산 서열을 설명한다. "X"로 특정된 아미노산은 임의의 아미노산을 나타내고, 바람직하게는 표 6에 표시되고 도 7a 및 7b에 제시된 서열의 EasD 서열 정렬의 적어도 하나의 아미노산 서열에서 동일한 위치를 갖는 아미노산을 나타낸다.
EasD 컨센서스 서열 (서열 152):
바람직하게는, 본 발명을 위해 설명된 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드의 EasD 활성은 다음 군으로부터 선택된 적어도 하나의 폴리펩티드 또는 적어도 하나의 폴리펩티드를 코딩하는 적어도 하나의 폴리뉴클레오티드의 활성에 의한 것이다:
a) 서열 4에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
b) 서열 4에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 152에 적어도 80%, 바람직하게는 적어도 81% 서열 동일성을 갖고, 보다 더 바람직하게는 도 7에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는 폴리펩티드,
c) 서열 53에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
d) 서열 53에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 152에 적어도 85%, 바람직하게는 적어도 86% 서열 동일성을 갖고, 보다 더 바람직하게는 도 7에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는 폴리펩티드, 및
e) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 d) 중 적어도 하나에 따른 폴리펩티드.
본 발명을 위해 설명된 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드 중의 임의의 하나의 EasH 활성은 바람직하게는 다음 군으로부터 선택된 적어도 하나의 폴리펩티드 또는 적어도 하나의 폴리뉴클레오티드의 활성에 의한 것이다:
a) 서열 156에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
b) 서열 8 및/또는 95에 적어도 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 156에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드 또는
c) 서열 8 및/또는 95에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 109 및/또는 157에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
e) 서열 109, 136 또는 144 및/또는 157에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는,
f) 서열 109, 136, 144 및/또는 157에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
g) 엄격한 조건 하에 서열 109, 136, 144 및/또는 157에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
h) a) 내지 g) 중 적어도 2개에 따른 폴리펩티드, 및
i) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 h) 중 적어도 하나에 따른 폴리펩티드.
EasH 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드는 원칙적으로 본원에 언급된 임의의 천연 맥각 알칼로이드 생산자로부터 얻을 수 있다. 한 실시양태에서, 폴리뉴클레오티드는 아스페르길루스 자포니카스(Aspergillus japonicas) 및 파에실로미세스 디바리카투스로 이루어진 군으로부터 선택되는 천연 맥각 알칼로이드 생산자로부터 얻어진다.
EasH 활성을 갖는 추가의 폴리펩티드는 문헌 [Stauffacher D. et al. (1969) Tetrahedron Vol.25: p5879-5887)]에 개시된 이포모에아 힐데브란티이 (Ipomoea hildebrandtii) 바트케 (Vatke))의 씨드에 포함된 내부 기생식물에 포함될 것이다.
바람직하게는, EasH 활성을 갖는 폴리펩티드는 EasH 컨센서스 서열을 포함한다. EasH 컨센서스 서열은 각각의 개별적인 아미노산이 관련 기술 분야에서 사용되는 단일 문자 아미노산 코드에 따라 특정되는 연속적인 아미노산 서열을 설명한다. "X"로 특정된 아미노산은 임의의 아미노산을 나타내고, 바람직하게는 서열 8 및 95에 의해 설명되고 도 8에 제시된 서열의 EasH 서열 정렬의 적어도 하나의 아미노산 서열에서 동일한 위치를 갖는 아미노산을 나타낸다.
EasH 컨센서스 서열 (서열 156):
바람직하게는, 본 발명을 위해 설명된 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드의 EasH 활성은 다음 군으로부터 선택된 적어도 하나의 폴리펩티드 또는 적어도 하나의 폴리펩티드를 코딩하는 적어도 하나의 폴리뉴클레오티드의 활성에 의한 것이다:
a) 서열 8에 적어도 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
b) 서열 8에 적어도 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 156에 적어도 90%, 바람직하게는 적어도 95% 서열 동일성을 갖고, 보다 더 바람직하게는 도 8에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는 폴리펩티드,
c) 서열 95에 적어도 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
d) 서열 95에 적어도 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 156에 적어도 90%, 바람직하게는 적어도 95% 서열 동일성을 갖고, 보다 더 바람직하게는 도 8에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는 폴리펩티드,
e) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 d) 중 적어도 하나에 따른 폴리펩티드.
본 발명을 위해 설명된 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드 중의 임의의 하나의 EasA 리덕타제 활성은 바람직하게는 다음 군으로부터 선택된 적어도 하나의 폴리펩티드 또는 적어도 하나의 폴리뉴클레오티드의 활성에 의한 것이다:
a) 서열 148, 149 및 150에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하고 서열 149의 아미노산 위치 18에 티로신을 갖는 폴리펩티드, 또는
b) 서열 193, 194 및 195에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하고 서열 194의 아미노산 위치 18에 티로신을 갖는 폴리펩티드, 또는
c) 서열 2 및/또는 31에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 2, 31, 32, 33, 34, 35, 36, 37, 38, 39 및/또는 40에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
e) 서열 103에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
f) 서열 130 및/또는 138에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는,
g) 서열 103, 130 및/또는 138에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는 (서열 목록에 포함된 상동체),
h) 엄격한 조건 하에 서열 103, 130 및/또는 138에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
i) a) 내지 h) 중 적어도 2개, 바람직하게는 적어도 b) 및 c)에 따른 폴리펩티드 및
j) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 i) 중 적어도 하나에 따른 폴리펩티드.
EasA 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드는 원칙적으로 본원에 언급된 임의의 천연 맥각 알칼로이드 생산자로부터 얻을 수 있다. 한 실시양태에서, 폴리뉴클레오티드는
아스페르길루스 테레우스, 발란시아 안드로포고니스, 발란시아 사이페리, 발란시아 오브텍타, 발란시아 필루아에포르미스, 클라비셉스 아프리카나, 클라비셉스 푸시포르미스, 클라비셉스 히르텔라, 스파셀리아 에리오클로아에, 스파셀리아 텍센시스, 스파셀리아 로벨레시이, 네오티포디움 종 Lp1, 네오티포디움 코에노피알룸, 네오티포디움 롤리이, 페니실리움 시트리눔, 페니실리움 콤뮨, 페니실리움 코릴로필룸, 페니실리움 크루스토숨, 페니실리움 펠루타눔 및 페니실리움 팔리탄스로 이루어지는 군, 또는
아스페르길루스 자포니쿠스, 아스페르길루스 니거, 아스페르길루스 오리자에, 아스페르길루스 니둘란스, 아스페르길루스 푸미가투스, 클라비셉스 푸르푸레아, 클라비셉스 푸시포르미스, 클라비셉스 파스팔리, 클라비셉스 지자니아에, 페니실리움 크리소게눔, 페니실리움 시트리눔, 페니실리움 코릴로피눔, 페니실리움 펠루타눔, 페니실리움 와크스마니이, 페니실리움 로쿠에포르티 및 파에실로미세스 디바리카투스로 이루어지는 군, 또는
아스페르길루스 자포니쿠스, 아스페르길루스 푸미가투스, 클라비셉스 푸르푸레아, 및 파에실로미세스 디바리카투스로 이루어지는 군, 또는
상기 군의 임의의 하나의 조합인 군으로 이루어지는 군
으로부터 선택된 천연 맥각 알칼로이드 생산자로부터 얻어진다.
바람직하게는, EasA 활성을 갖는 적어도 하나의 폴리펩티드는 표 7에 나열된 서열, 또는 상기 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리펩티드로부터 선택된다.
<표 7>
바람직하게는, EasA 활성을 갖는 폴리펩티드는 아래에서 설명되는 적어도 하나의 EasA 시그너처 서열을 포함한다. 바람직하게는, 이것은 적어도 2개의 EasA 시그너처 서열을 포함하고, 보다 더 바람직하게는 3개의 모든 EasA 시그너처 서열을 포함한다.
EasA 시그너처 및 컨센서스 서열은 각각의 개별적인 아미노산이 관련 기술 분야에서 사용되는 단일 문자 아미노산 코드에 따라 특정되는 연속적인 아미노산 서열을 설명한다. "X"로 특정된 아미노산은 임의의 아미노산을 나타내고, 바람직하게는 표 7에 표시되고 도 9a 내지 9d에 제시된 서열의 EasA 서열 정렬의 적어도 하나의 아미노산 서열에서 동일한 위치를 갖는 아미노산을 나타낸다. 보다 더 바람직하게는, EasA 활성을 갖는 폴리펩티드는 도 9a, 9b 및 9c에 흑색 막대로 표시된 전체 아미노산 서열을 포함하고, 도 9b에 흑색 화살표로 표시된 아미노산 위치에 페닐알라닌 (F) 또는 티로신 (Y)을 포함한다.
EasA 시그너처 서열 1.1 (서열 148):
EasA 시그너처 서열 2.1 (서열 149):
EasA 시그너처 서열 3.1 (서열 150):
EasA 시그너처 서열 1.2 (서열 193):
EasA 시그너처 서열 2.2 (서열 194):
EasA 시그너처 서열 3.2 (서열 195):
바람직하게는, 본 발명을 위해 설명된 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드의 EasA 활성은 다음 군으로부터 선택된 적어도 하나의 폴리펩티드 또는 적어도 하나의 폴리펩티드를 코딩하는 적어도 하나의 폴리뉴클레오티드의 활성에 의한 것이다:
a) 서열 2에 적어도 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 149의 아미노산 위치 18에 티로신을 갖는 아미노산 서열을 포함하는 폴리펩티드,
b) 서열 2에 적어도 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 148, 149 및 150에 적어도 95%, 바람직하게는 100% 서열 동일성을 갖고 서열 149의 아미노산 위치 18에 티로신을 갖는 아미노산 서열을 포함하는 폴리펩티드,
c) 서열 31에 적어도 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 149의 아미노산 위치 18에 티로신을 갖는 아미노산 서열을 포함하는 폴리펩티드,
d) 서열 31에 적어도 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 148, 149 및 150에 적어도 95%, 바람직하게는 100% 서열 동일성을 갖고 서열 149의 아미노산 위치 18에 티로신을 갖는 아미노산 서열을 포함하는 폴리펩티드, 및
e) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 d) 중 적어도 하나에 따른 폴리펩티드.
EasA 리덕타제 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드는 바람직하게는 아스페르길루스 자포니쿠스, 아스페르길루스 푸미가투스, 아스페르길루스 플라부스, 아스페르길루스 테레우스, 클라비셉스 히르텔라, 클라비셉스 아프리카나, 페니실리움 크리소게눔, 및 파에실로미세스 디바리카투스로 이루어지는 군으로부터 선택된 천연 맥각 알칼로이드 생산자 유기체의 군으로부터 얻어진다.
본 발명을 위해 설명된 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드 중의 임의의 하나의 EasA 이소머라제 활성은 바람직하게는 다음 군으로부터 선택된 적어도 하나의 폴리펩티드 또는 적어도 하나의 폴리뉴클레오티드의 활성에 의한 것이다:
a) 서열 148, 149 및 150에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 149의 아미노산 위치 18에 페닐알라닌을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
b) 서열 193, 194 및 195에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 194의 아미노산 위치 18에 페닐알라닌을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
c) 서열 2 및/또는 31에 적어도 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 2, 31, 32, 33, 34, 35, 36, 37, 38, 39 및/또는 40에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
e) 서열 103에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
f) 서열 130 및/또는 138에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는,
g) 서열 103, 130 및/또는 138에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
h) 엄격한 조건 하에 서열 103, 130 및/또는 138에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
i) a) 내지 h) 중 적어도 2개에 따른 폴리펩티드, 및
j) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 i) 중 적어도 하나에 따른 폴리펩티드.
바람직하게는, 본 발명을 위해 설명된 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드의 EasA 활성은 다음 군으로부터 선택된 적어도 하나의 폴리펩티드 또는 적어도 하나의 폴리펩티드를 코딩하는 적어도 하나의 폴리뉴클레오티드의 활성에 의한 것이다:
a) 서열 2에 적어도 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 149의 아미노산 위치 18에 페닐알라닌을 갖는 아미노산 서열을 포함하는 폴리펩티드,
b) 서열 2에 적어도 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 148, 149 및 150에 적어도 95%, 바람직하게는 100% 서열 동일성을 갖고 서열 149의 아미노산 위치 18에 페닐알라닌을 갖는 아미노산 서열을 포함하는 폴리펩티드,
c) 서열 31에 적어도 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 149의 아미노산 위치 18에 페닐알라닌을 갖는 아미노산 서열을 포함하는 폴리펩티드,
d) 서열 31에 적어도 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 148, 149 및 150에 적어도 95%, 바람직하게는 100% 서열 동일성을 갖고 서열 149의 아미노산 위치 18에 페닐알라닌을 갖는 아미노산 서열을 포함하는 폴리펩티드, 및
e) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 d) 중 적어도 하나에 따른 폴리펩티드.
EasA 이소머라제 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드는 바람직하게는 아스페르길루스 테레우스, 발란시아 안드로포고니스, 발란시아 사이페리, 발란시아 오브텍타, 발란시아 필루아에포르미스, 클라비셉스 푸르푸레아, 클라비셉스 히르텔라, 클라비셉스 푸시포르미스, 클라비셉스 파스팔리, 클라비셉스 지자니아에, 스파셀리아 에리오클로아에, 스파셀리아 텍센시스, 스파셀리아 로벨레시이, 네오티포디움 종 Lp1, 네오티포디움 코에노피알룸, 네오티포디움 롤리이, 페니실리움 시트리눔, 페니실리움 코릴로필룸, 페니실리움 펠루타눔 및 페리글란둘라 이포모에아에로 이루어지는 군, 또는 클라비셉스 푸르푸레아, 클라비셉스 푸시포르미스, 네오티포디움 롤리이 및 페리글란둘라 이포모에아에로 이루어지는 군으로부터 선택된 천연 맥각 알칼로이드 생산자 유기체의 군으로부터 얻어진다.
본 발명을 위해 설명된 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드 중의 임의의 하나의 EasG 활성은 바람직하게는 다음 군으로부터 선택된 적어도 하나의 폴리펩티드 또는 적어도 하나의 폴리뉴클레오티드의 활성에 의한 것이다:
a) 서열 155 및/또는 183에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고, 바람직하게는 서열 183에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
b) 서열 7 및/또는 86에 적어도 39% 40%, 42%, 44%, 46%, 48%, 50%, 52%, 54%, 56%. 58%, 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
c) 서열 7, 86, 87, 88, 89, 90, 91, 92, 93 및/또는 94에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
d) 서열 108에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드,
e) 서열 135 및/또는 143에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드,
f) 서열 108, 135 및/또는 143에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드,
g) 엄격한 조건 하에 서열 108, 135 및/또는 143에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드,
h) a) 내지 g) 중 적어도 2개, 바람직하게는 적어도 a) 및 b)에 따른 폴리펩티드, 및
i) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 h) 중 적어도 하나에 따른 폴리펩티드.
EasG 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드는 원칙적으로 본원에 언급된 임의의 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있다. 한 실시양태에서, 폴리뉴클레오티드는
아스페르길루스 테레우스, 발란시아 안드로포고니스, 발란시아 사이페리, 발란시아 오브텍타, 발란시아 필루아에포르미스, 클라비셉스 아프리카나, 클라비셉스 푸시포르미스, 클라비셉스 히르텔라, 스파셀리아 에리오클로아에, 스파셀리아 텍센시스, 스파셀리아 로벨레시이, 네오티포디움 종 Lp1, 네오티포디움 코에노피알룸, 네오티포디움 롤리이, 페니실리움 시트리눔, 페니실리움 콤뮨, 페니실리움 코릴로필룸, 페니실리움 크루스토숨, 페니실리움 펠루타눔 및 페니실리움 팔리탄스로 이루어지는 군, 또는
아스페르길루스 자포니쿠스, 아스페르길루스 니거, 아스페르길루스 오리자에, 아스페르길루스 니둘란스, 아스페르길루스 푸미가투스, 클라비셉스 푸르푸레아, 클라비셉스 푸시포르미스, 클라비셉스 파스팔리, 클라비셉스 지자니아에, 페니실리움 크리소게눔, 페니실리움 시트리눔, 페니실리움 코릴로피눔, 페니실리움 펠루타눔, 페니실리움 와크스마니이, 페니실리움 로쿠에포르티 및 파에실로미세스 디바리카투스로 이루어지는 군, 또는
에피클로에 페스투카에, 에피클로에 글리세리아에, 메타리지움 아크리둠, 메타리지움 로베르트시이 및 트리코피톤 베루코숨으로 이루어지는 군, 또는
아스페르길루스 자포니쿠스, 아스페르길루스 푸미가투스, 클라비셉스 푸르푸레아, 및 파에실로미세스 디바리카투스로 이루어지는 군, 또는
상기 군의 임의의 하나의 조합인 군으로 이루어지는 군
으로부터 선택된 천연 맥각 알칼로이드 생산자로부터 얻어진다.
바람직하게는, EasG 활성을 갖는 적어도 하나의 폴리펩티드는 표 8에 나열된 서열, 또는 상기 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리펩티드로부터 선택된다.
<표 8>
바람직하게는, EasG 활성을 갖는 폴리펩티드는 EasG 컨센서스 서열을 포함한다. EasG 컨센서스 서열은 각각의 개별적인 아미노산이 관련 기술 분야에서 사용되는 단일 문자 아미노산 코드에 따라 특정되는 연속적인 아미노산 서열을 설명한다. "X"로 특정된 아미노산은 임의의 아미노산을 나타내고, 바람직하게는 표 8에 표시되고 도 10a 내지 10b에 제시된 서열의 EasG 서열 정렬의 적어도 하나의 아미노산 서열에서 동일한 위치를 갖는 아미노산을 나타낸다.
EasG 컨센서스 서열 (서열 155):
바람직하게는, 본 발명을 위해 설명된 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드의 EasG 활성은 다음 군으로부터 선택된 적어도 하나의 폴리펩티드 또는 적어도 하나의 폴리펩티드를 코딩하는 적어도 하나의 폴리뉴클레오티드의 활성에 의한 것이다:
a) 서열 7에 적어도 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
b) 서열 7에 적어도 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 183에 적어도 90%, 바람직하게는 적어도 95% 서열 동일성을 갖고, 보다 더 바람직하게는 도 10에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는 폴리펩티드,
c) 서열 86에 적어도 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드,
d) 서열 86에 적어도 60%, 62%, 64%, 66%, 68%, 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 183에 적어도 90%, 바람직하게는 적어도 95% 서열 동일성을 갖고, 보다 더 바람직하게는 도 10에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는 폴리펩티드, 및
e) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 d) 중 적어도 하나에 따른 폴리펩티드.
본원에서 설명되는 모든 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체는 바람직하게는 상기 설명한 트립토판 및/또는 DMAPP의 확대된 세포 내부 공급원 및/또는 Me-DMAT의 확대된 세포 내부 공급원을 포함한다. 확대된 세포 내부 공급원을 제공하기 위해, 대부분의 경우에 각각의 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체의 내인성 유전자를 탈조절하는 것이 필요할 것이다.
재조합 미생물이 효모, 바람직하게는 사카로미세스 종으로부터 선택되는 경우에, 보다 더 바람직하게는 재조합 미생물이 사카로미세스 세레비지아에인 경우에, 본 발명의 하나의 바람직한 실시양태는 서열 9에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 상기 재조합 미생물의 내인성 폴리펩티드를 하향조절함으로써 ERG9 활성 (EC 2.5.1.21)을 하향조절하는 것이다.
별법으로 또는 그에 추가로, 효모, 바람직하게는 사카로미세스 종, 보다 더 바람직하게는 사카로미세스 세레비지아에 세포의 DMAPP의 내부 공급원은 ERG20 활성을 갖는 폴리펩티드 (EC 2.5.1.1/2.5.1.10), 특히 서열 10에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드를 하향조절함으로써 확대될 수 있다.
또한, 효모, 바람직하게는 사카로미세스 종, 보다 더 바람직하게는 사카로미세스 세레비지아에 세포의 DMAPP의 내부 공급원은 예를 들어 서열 11에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드에 의한 것인 HMG-CoA 리덕타제 활성을 조정함으로써 상기 유기체의 메발로네이트 경로의 유전자를 상향조절함으로써 확대될 수 있다.
세포 내의 클라빈-유형 알칼로이드의 전구체의 내부 공급원을 확대하는 또 다른 방법은 Me-DMAT의 배지 내로의 외수송을 제한하는 것이다. 이것은 메발로네이트 경로의 세포 내인성 폴리펩티드의 활성을 상향조절함으로써 달성될 수 있다. 재조합 미생물이 효모, 바람직하게는 사카로미세스 종, 보다 더 바람직하게는 사카로미세스 세레비지아에 세포인 경우에, 이것은 서열 12, 13, 14, 15 또는 16에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 하나 이상의 폴리펩티드의 활성을 하향조절함으로써 달성될 수 있다.
본 발명은 새로운 맥각 알칼로이드 생산자 유기체, 특히 맥각 알칼로이드를 생산하는 능력을 갖지 않는 유기체를 본 발명의 폴리뉴클레오티드, 발현 카세트 및/또는 벡터로 형질전환시킴으로써 클라빈-유형 알칼로이드를 생산할 수 있는 새로운 맥각 알칼로이드 생산자 유기체를 생성할 가능성을 제공한다.
많은 경우에, 각각의 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체에서 재조합 발현된 폴리펩티드의 적합한 발현, 폴딩 및 안정성을 지지하는 유전자의 과다발현에 의해 상기 기능을 지지하는 것이 또한 유용하다.
상기 기능을 지지할 수 있는 유전자는 예를 들어, ScIdi1 (서열 158), ScPdi1 (서열 166) 및 ScFad1 (서열 167)이다.
따라서, 추가의 실시양태는 그에 추가로 서열 158, 166 또는 167에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 적어도 하나의 폴리펩티드의 상향조절된 활성을 포함하는, 바람직하게는 서열 166 및 167에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 적어도 하나의 폴리펩티드의 상향조절된 활성을 갖는, 본원에서 설명되는 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I의 생산을 위한 폴리펩티드를 발현하는 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체이다.
한 실시양태에서 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체는 시클로클라빈의 생산을 위해 본원에서 설명되는 상향조절된 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 및 EasG 활성을 갖는 및/또는 적어도 하나의 페스투클라빈 또는 아그로클라빈의 생산을 위해 본원에서 설명되는 상향조절된 DmaW, EasF, EasE, EasC, EasD, EasA 및 EasG 활성을 갖는 효모, 바람직하게는 에스. 세레비지아에(S. cerevisiaee)이다. 바람직하게는, 상기 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체는 또한 상기 설명한 DMAPP의 확대된 내부 공급원을 갖는다.
본 발명의 추가의 실시양태는 시클로클라빈의 생산을 위해 본원에서 설명되는 상향조절된 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 및 EasG 활성을 갖는 및/또는 적어도 하나의 페스투클라빈 또는 아그로클라빈의 생산을 위해 본원에서 설명되는 상향조절된 DmaW, EasF, EasE, EasC, EasD, EasA 및 EasG 활성을 갖는 및 서열 158, 166 및/또는 167에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 적어도 하나의 폴리펩티드의 상향조절된 활성을 갖는, 바람직하게는 서열 166 및 167에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 적어도 하나의 폴리펩티드의 상향조절된 활성을 갖는 재조합 에스. 세레비지아에이다.
바람직하게는, 시클로클라빈, 페스투클라빈 및/또는 아그로클라빈의 생산을 위해 본원에서 설명되는 적어도 상향조절된 EasE 활성을 갖는 에스. 세레비지아에는 또한 서열 187에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 적어도 하나의 폴리펩티드의 상향조절된 활성을 갖고, 바람직하게는, 서열 187에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드는 서열 186에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 뉴클레오티드 서열을 갖는 폴리뉴클레오티드에 의해 코딩된다.
바람직하게는, 상기 재조합 에스. 세레비지아에는 또한 바람직하게는 서열 9 또는 10에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 내인성 폴리펩티드를 하향조절함으로써, 또는 서열 9에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 내인성 폴리펩티드를 하향조절함으로써 및 서열 10에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 내인성 폴리펩티드를 하향조절함으로써 상기 설명한 DMAPP의 확대된 내부 공급원을 갖는다.
따라서, 본 발명의 추가의 실시양태는
a) 시클로클라빈,
b) 페스투클라빈,
c) 아그로클라빈,
d) 카노클라빈 알데히드, 및
e) 카노클라빈 I
의 화합물의 군으로부터 선택되는 적어도 하나의 화합물을 포함하는 재조합 미생물이고, 여기서 재조합 미생물은 천연 맥각 알칼로이드 생산자 유기체가 아니다.
바람직하게는, 상기 재조합 미생물은 천연 맥각 알칼로이드 생산자 유기체가 아닌 박테리아, 효모, 방선균, 또는 사상 진균이다. 상기 사상 진균은 자낭균, 불완전균류, 또는 담자균일 수 있다.
본 발명의 숙주 세포로서 사용되는 바람직한 박테리아는 에스케리키아 콜라이 및 바실러스 서브틸리스로 이루어지는 군으로부터 선택된다.
바람직한 재조합 미생물은 다음으로 이루어지는 군으로부터 선택된다:
바람직한 효모는 클루이베로미세스, 사카로미세스, 쉬조사카로미세스, 야로위아 및 피키아의 군으로부터 선택된다.
클루이베로미세스 락티스, 사카로미세스 세레비지아에, 쉬조사카로미세스 폼베, 야로위아 리폴리티카 및 피키아 스티피테스로 이루어지는 군으로부터 선택된 재조합 미생물이 보다 바람직하다.
보다 더 바람직한 재조합 미생물은 사카로미세스 세레비지아에이다.
상기 유기체의 상이한 균주, 예를 들어 공공 균주 기탁기관에 기탁된 균주는 관련 기술 분야의 통상의 기술자에게 쉽게 이용가능하다. 사카로미세스 세레비지아에의 적합한 균주는 예를 들어 BY4742, cGY 1585, 에탄올 레드, CEN.PK 111-61A, FY1679-06C, BMA64 (W303), SEY 6210이고, 이로 제한되지 않는다.
재조합 미생물이 박테리아, 특히 이. 콜라이인 경우에, 메발로네이트 경로의 유전자의 과다발현이 유용할 수 있다. 메발로네이트 경로의 상향조절된 유전자를 포함하는 이. 콜라이는 관련 기술 분야에 알려져 있고, 예를 들어 US7172886, US7192751, US7667017, US7622282, US7736882, US7622283, US7915026, 및 US8288147에 기재되어 있다.
시클로클라빈을 포함하는 재조합 미생물은 바람직하게는 각각의 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 및 EasG 활성, 보다 바람직하게는 각각의 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 리덕타제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함할 것이다.
페스투클라빈을 포함하는 재조합 미생물은 바람직하게는 각각의 DmaW, EasF, EasE, EasC, EasD, EasA 리덕타제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함할 것이다.
아그로클라빈을 포함하는 재조합 미생물은 바람직하게는 각각의 DmaW, EasF, EasE, EasC, EasD, EasA 이소머라제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함할 것이다.
카노클라빈 알데히드를 포함하는 재조합 미생물은 바람직하게는 각각의 DmaW, EasF, EasE, EasC 및 EasD 활성에 대한 적어도 하나의 폴리펩티드를 포함할 것이다.
카노클라빈 I을 포함하는 재조합 미생물은 바람직하게는 각각의 DmaW, EasF, EasE 및 EasC 활성에 대한 적어도 하나의 폴리펩티드를 포함할 것이다.
Me-DMAT를 포함하는 재조합 미생물은 바람직하게는 각각의 DmaW 및 EasF 활성에 대한 적어도 하나의 폴리펩티드를 포함할 것이다.
DMAT를 포함하는 재조합 미생물은 바람직하게는 DmaW 활성에 대한 적어도 하나의 폴리펩티드를 포함할 것이다.
또한, 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드, 또는 카노클라빈 I 또는 상기 화합물의 적어도 2개의 조합물을 포함하지만, 트립토판 및 DMAPP의 세포 내부 풀로부터 각각의 화합물 또는 화합물들을 생산하기 위해 필요한 것보다 작은 활성을 갖는 재조합 미생물을 생산할 수 있다. 상실된 활성은 재조합 미생물에게 다른 수단에 의해 필요한 전구체를 제공함으로써, 예를 들어 전구체를 성장 배지에 제공함으로써 보충될 수 있다.
따라서, 본 발명의 한 실시양태는 DMAT가 제공되고 각각의 EasF, EasE, EasC, EasD, EasH, EasA 및 EasG 활성, 보다 바람직하게는 각각의 EasF, EasE, EasC, EasD, EasH, EasA 리덕타제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 시클로클라빈을 포함하는 재조합 미생물이다.
추가의 실시양태는 Me-DMAT가 제공되고 각각의 EasE, EasC, EasD, EasH, EasA 및 EasG 활성, 보다 바람직하게는 각각의 EasE, EasC, EasD, EasH, EasA 리덕타제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 시클로클라빈을 포함하는 재조합 미생물이다.
또 다른 추가의 실시양태는 카노클라빈 I이 제공되고 각각의 EasD, EasH, EasA 및 EasG 활성, 보다 바람직하게는 각각의 EasD, EasH, EasA 리덕타제 및 EasG 활성을 제공하는 적어도 하나의 폴리펩티드를 포함하는, 시클로클라빈을 포함하는 재조합 미생물이다.
추가의 실시양태는 DMAT가 제공되고 각각의 EasF, EasE, EasC, EasD, EasA 리덕타제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 페스투클라빈을 포함하는 재조합 미생물이다.
Me-DMAT가 제공되고 각각의 EasE, EasC, EasD, EasA 리덕타제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 페스투클라빈을 포함하는 재조합 미생물이 제시된다.
카노클라빈 I이 제공되고 각각의 EasD, EasA 리덕타제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 페스투클라빈을 포함하는 재조합 미생물이 제시된다.
카노클라빈 알데히드가 제공되고 각각의 EasA 리덕타제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 페스투클라빈을 포함하는 재조합 미생물이 제시된다.
DMAT가 제공되고 각각의 EasF, EasE, EasC, EasD, EasA 이소머라제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 아그로클라빈을 포함하는 재조합 미생물이 제시된다.
Me-DMAT가 제공되고 각각의 EasE, EasC, EasD, EasA 이소머라제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 아그로클라빈을 포함하는 재조합 미생물이 제시된다.
카노클라빈 I이 제공되고 각각의 EasD, EasA 이소머라제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 아그로클라빈을 포함하는 재조합 미생물이 제시된다.
카노클라빈 알데히드가 제공되고 각각의 EasA 이소머라제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 아그로클라빈을 포함하는 재조합 미생물이 제시된다.
DMAT가 제공되고 각각의 EasF, EasE, EasC 및 EasD 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 카노클라빈 알데히드를 포함하는 재조합 미생물이 제시된다.
Me-DMAT가 제공되고 각각의 EasE, EasC 및 EasD 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 카노클라빈 알데히드를 포함하는 재조합 미생물이 제시된다.
카노클라빈 I이 제공되고 EasD 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 카노클라빈 알데히드를 포함하는 재조합 미생물이 제시된다.
DMAT가 제공되고 각각의 EasF, EasE 및 EasC 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 카노클라빈 I을 포함하는 재조합 미생물이 제시된다.
Me-DMAT가 제공되고 EasE 및 EasC 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 카노클라빈 I을 포함하는 재조합 미생물이 제시된다.
세포가 갖는 추가의 활성에 따라, 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 또는 상기 화합물의 적어도 2개의 조합물의 함량은 적거나 많을 수 있다.
예를 들어, 아그로클라빈을 포함하지만 생성물, 예를 들어 엘리모클라빈 또는 세토클라빈을 생산하기 위해 추출물로서 아그로클라빈을 이용하는 활성을 또한 포함하거나, 또는 아그로클라빈의 주위 배지 내로의 외수송을 용이하게 하는 활성을 포함하는 재조합 미생물은 낮은 함량의 아그로클라빈을 포함할 수 있다.
유사하게, 페스투클라빈을 포함하지만 생성물, 예를 들어 푸미클라빈 B, A 또는 C를 생산하거나 또는 디히드로맥각 알칼로이드를 생산하기 위해 추출물로서 페스투클라빈을 이용하는 활성을 또한 포함하거나, 또는 페스투클라빈의 주위 배지 내로의 외수송을 용이하게 하는 활성을 포함하는 재조합 미생물은 낮은 함량의 페스투클라빈을 포함할 수 있다.
카노클라빈 알데히드를 포함하지만 생성물, 예를 들어 페스투클라빈 또는 아그로클라빈을 생산하기 위해 추출물로서 카노클라빈 알데히드를 이용하는 활성을 또한 포함하거나, 또는 카노클라빈 알데히드의 주위 배지 내로의 외수송을 용이하게 하는 활성을 포함하는 재조합 미생물은 낮은 함량의 카노클라빈 알데히드를 포함할 수 있다.
카노클라빈 I을 포함하지만 생성물, 예를 들어 시클로클라빈, 카노클라빈 알데히드 또는 6,7-세콜리에르긴을 생산하기 위해 추출물로서 카노클라빈 I을 이용하는 활성을 또한 포함하거나, 또는 카노클라빈 I의 주위 배지 내로의 외수송을 용이하게 하는 활성을 포함하는 재조합 미생물은 낮은 함량의 카노클라빈 I을 포함할 수 있다.
그러나, 각각의 화합물을 추출물로서 이용하거나 또는 상기 화합물의 주위 배지 내로의 외수송을 용이하게 하는 활성이 하향조절되는 경우에, 재조합 미생물 내의 화합물의 함량은 높을 수 있다.
본 발명은 그들의 비-재조합 형태에 비해 하나 이상의 맥각 알칼로이드의 향상된 생산을 보이는 재조합 천연 맥각 알칼로이드 생산자 유기체를 생성할 가능성을 추가로 제공한다. 또한, 맥각 알칼로이드 생산을 위한 내인성 유전자를 하향조절함으로써 또는 본원에 개시된 하나 이상의 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 및 EasG 활성을 도입함으로써, 천연 맥각 알칼로이드 생산자에 의해 대체로 생산되는 맥각 알칼로이드 유형의 변화, 특히 천연 맥각 알칼로이드 생산자에 의해 생산되는 클라빈-유형 알칼로이드의 생산의 변화도 가능하다.
하나 이상의 맥각 알칼로이드의 향상된 생산은 바람직하게는 본 발명의 폴리뉴클레오티드의 발현이 결여되고 바람직하게는 재조합 천연 맥각 알칼로이드 생산자 유기체를 구축하기 위해 사용된 동일한 종 및 바람직하게는 동일한 균주의 야생형 유기체, 즉, 비-재조합 유기체이고 대조군 유기체로서 사용될 때 재조합 천연 맥각 알칼로이드 생산자 유기체와 동일한 조건 하에 성장하는 대조군 유기체에 비해 통계적으로 유의하다.
증가의 유의성 여부는 예를 들어 스튜던트 (Student) t-시험을 비롯하여 관련 기술 분야에 잘 알려진 통계적인 시험에 의해 결정될 수 있다. 보다 바람직하게는, 증가는 상기 대조군 유기체에 비해 각각의 맥각 알칼로이드의 양의 적어도 5%, 적어도 10%, 적어도 15%, 적어도 20% 또는 적어도 30%의 증가이다. 맥각 알칼로이드의 양을 측정하기 위한 적합한 검정은 첨부되는 실시예에서 설명되고, 관련 기술 분야에 알려져 있다.
따라서, 본 발명의 추가의 실시양태는 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및/또는 카노클라빈 I의 화합물의 군으로부터 선택되는 적어도 하나의 화합물을 포함하는 재조합 천연 맥각 알칼로이드 생산자 유기체이고, 여기서 재조합 천연 맥각 알칼로이드 생산자 유기체는 자연에서 상기 화합물을 생산하지 않는 천연 맥각 알칼로이드 생산자 유기체로부터의 것이다.
그에 추가로, 본 발명의 폴리펩티드 및 폴리뉴클레오티드는 또한 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 및 EasG 활성의 군으로부터 선택된 적어도 하나의 상향조절된 또는 적어도 하나의 하향조절된, 또는 적어도 하나의 상향조절된 및 적어도 하나의 하향조절된 활성을 갖는 재조합 천연 맥각 알칼로이드 생산자 유기체를 생성함으로써 천연 맥각 알칼로이드 생산자 유기체의 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및/또는 카노클라빈 I의 생산을 향상시키기 위해 사용될 수 있다.
따라서, 본 발명의 추가의 실시양태는 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및 카노클라빈 I의 군으로부터 선택되는 적어도 하나의 화합물을 동일한 조건 하에 성장할 때 비-재조합 야생형 유기체에 비해 보다 많은 양으로 포함하고/하거나 생산하는 재조합 천연 맥각 알칼로이드 생산자 유기체이다.
비교를 위해 사용된 각각의 야생형 유기체는 바람직하게는 각각의 재조합 천연 맥각 알칼로이드 생산자 유기체를 생성하기 위해 사용된 것과 동일한 종의 것이고, 바람직하게는 동일한 균주의 것일 것이다.
시클로클라빈을 포함하거나 시클로클라빈을 생산하는 재조합 천연 맥각 알칼로이드 생산자 유기체는 적어도 하나의 상향조절된 EasH 활성을 포함할 것이고, 바람직하게는 적어도 하나의 상향조절된 EasH 활성 및 적어도 하나의 DmaW, EasF, EasE, EasC, EasD, EasA 및/또는 EasG 활성을 포함할 것이고, 보다 바람직하게는 적어도 하나의 상향조절된 EasH 활성 및 적어도 하나의 DmaW, EasF, EasE, EasC, EasD, EasA 리덕타제 및/또는 EasG 활성을 포함할 것이고, 보다 더 바람직하게는 적어도 하나의 상향조절된 EasH 활성 및 적어도 하나의 상향조절된 DmaW, EasF, EasE, EasC, EasD, EasA 리덕타제 및/또는 EasG 활성 및 적어도 하나의 하향조절된 EasA 이소머라제 활성을 포함할 것이다.
페스투클라빈을 포함하거나 페스투클라빈을 생산하는 재조합 천연 맥각 알칼로이드 생산자 유기체는 적어도 하나의 상향조절된 DmaW, EasF, EasE, EasC, EasD, EasA 리덕타제 또는 EasG 활성, 바람직하게는 적어도 하나의 상향조절된 DmaW, EasF, EasE, EasC, EasD, EasA 리덕타제 또는 EasG 활성 및 적어도 하나의 하향조절된 EasA 이소머라제 및/또는 EasH 활성을 포함할 것이다.
아그로클라빈을 포함하거나 아그로클라빈을 생산하는 재조합 천연 맥각 알칼로이드 생산자 유기체는 적어도 하나의 상향조절된 DmaW, EasF, EasE, EasC, EasD, EasA 이소머라제 및/또는 EasG 활성, 바람직하게는 적어도 하나의 상향조절된 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 이소머라제 및/또는 EasG 활성 및 적어도 하나의 하향조절된 EasA 리덕타제 및/또는 EasH 활성을 포함할 것이다.
카노클라빈 알데히드를 포함하거나 카노클라빈 알데히드를 생산하는 재조합 천연 맥각 알칼로이드 생산자 유기체는 적어도 하나의 상향조절된 DmaW, EasF, EasE, EasC 및/또는 EasD 활성 또는 적어도 하나의 하향조절된 EasA, EasG 및/또는 EasH 활성을 포함할 것이고, 바람직하게는 적어도 하나의 상향조절된 DmaW, EasF, EasE, EasC 및/또는 EasD 활성 및 적어도 하나의 하향조절된 EasA, EasG 및/또는 EasH 활성을 포함할 것이다.
카노클라빈 I을 포함하거나 카노클라빈 I을 생산하는 재조합 천연 맥각 알칼로이드 생산자 유기체는 적어도 하나의 상향조절된 DmaW, EasF, EasE 및/또는 EasC 활성, 또는 적어도 하나의 하향조절된 EasD, EasA, EasG 및/또는 EasH 활성을 포함할 것이거나, 또는 적어도 하나의 상향조절된 DmaW, EasF, EasE 및/또는 EasC 활성 및 적어도 하나의 하향조절된 EasD, EasA, EasG 및/또는 EasH 활성을 포함할 것이다.
시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드, 카노클라빈 I 또는 상기 화합물의 적어도 2개의 조합물을 포함하고/하거나 생산하지만, 트립토판 및 DMAPP의 세포 내부 풀로부터 각각의 화합물 또는 화합물들을 생산하기 위해 필요한 것보다 작은 활성을 갖는 재조합 천연 맥각 알칼로이드 생산자 유기체를 생산할 수 있다. 상실된 활성은 재조합 천연 맥각 알칼로이드 생산자 유기체에게 다른 수단에 의해 필요한 전구체를 제공함으로써, 예를 들어 전구체를 성장 배지에 제공함으로써 보충될 수 있다.
따라서, 본 발명의 한 실시양태는 DMAT가 제공되고 각각의 EasF, EasE, EasC, EasD, EasH, EasA 및 EasG 활성, 보다 바람직하게는 각각의 EasF, EasE, EasC, EasD, EasH, EasA 리덕타제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 시클로클라빈을 포함하는 재조합 천연 맥각 알칼로이드 생산자 유기체이다.
추가의 실시양태는 Me-DMAT가 제공되고 각각의 EasE, EasC, EasD, EasH, EasA 및 EasG 활성, 보다 바람직하게는 각각의 EasE, EasC, EasD, EasH, EasA 리덕타제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 시클로클라빈을 포함하는 재조합 천연 맥각 알칼로이드 생산자 유기체이다.
또 다른 추가의 실시양태는 카노클라빈 I이 제공되고 각각의 EasD, EasH, EasA 및 EasG 활성, 보다 바람직하게는 각각의 EasD, EasH, EasA 리덕타제 및 EasG 활성을 제공하는 적어도 하나의 폴리펩티드를 포함하는, 시클로클라빈을 포함하는 재조합 천연 맥각 알칼로이드 생산자 유기체이다.
추가의 실시양태는 DMAT가 제공되고 각각의 EasF, EasE, EasC, EasD, EasA 리덕타제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 페스투클라빈을 포함하는 재조합 천연 맥각 알칼로이드 생산자 유기체이다.
Me-DMAT가 제공되고 각각의 EasE, EasC, EasD, EasA 리덕타제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 페스투클라빈을 포함하는 재조합 천연 맥각 알칼로이드 생산자 유기체가 제시된다.
카노클라빈 I이 제공되고 각각의 EasD, EasA 리덕타제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 페스투클라빈을 포함하는 재조합 천연 맥각 알칼로이드 생산자 유기체가 제시된다.
카노클라빈 알데히드가 제공되고 각각의 EasA 리덕타제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 페스투클라빈을 포함하는 재조합 천연 맥각 알칼로이드 생산자 유기체가 제시된다.
DMAT가 제공되고 각각의 EasF, EasE, EasC, EasD, EasA 이소머라제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 아그로클라빈을 포함하는 재조합 천연 맥각 알칼로이드 생산자 유기체가 제시된다.
Me-DMAT가 제공되고 각각의 EasE, EasC, EasD, EasA 이소머라제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 아그로클라빈을 포함하는 재조합 천연 맥각 알칼로이드 생산자 유기체가 제시된다.
카노클라빈 I이 제공되고 각각의 EasD, EasA 이소머라제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 아그로클라빈을 포함하는 재조합 천연 맥각 알칼로이드 생산자 유기체가 제시된다.
카노클라빈 알데히드가 제공되고 각각의 EasA 이소머라제 및 EasG 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 아그로클라빈을 포함하는 재조합 천연 맥각 알칼로이드 생산자 유기체가 제시된다.
DMAT가 제공되고 각각의 EasF, EasE, EasC 및 EasD 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 카노클라빈 알데히드를 포함하는 재조합 천연 맥각 알칼로이드 생산자 유기체가 제시된다.
Me-DMAT가 제공되고 각각의 EasE, EasC 및 EasD 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 카노클라빈 알데히드를 포함하는 재조합 천연 맥각 알칼로이드 생산자 유기체가 제시된다.
카노클라빈 I이 제공되고 EasD 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 카노클라빈 알데히드를 포함하는 재조합 천연 맥각 알칼로이드 생산자 유기체가 제시된다.
DMAT가 제공되고 각각의 EasF, EasE 및 EasC 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 카노클라빈 I을 포함하는 재조합 천연 맥각 알칼로이드 생산자 유기체가 제시된다.
Me-DMAT가 제공되고 EasE 및 EasC 활성을 위한 적어도 하나의 폴리펩티드를 포함하는, 카노클라빈 I을 포함하는 재조합 천연 맥각 알칼로이드 생산자 유기체가 제시된다.
바람직하게는, 재조합 천연 맥각 알칼로이드 생산자 유기체는
속 아스페르길루스, 예컨대 아스페르길루스 자포니쿠스, 아스페르길루스 니거, 아스페르길루스 오리자에, 아스페르길루스 니둘란스, 아스페르길루스 푸미가투스, 아스페르길루스 아쿨레아투스, 아스페르길루스 카에시엘루스, 아스페르길루스 칸디두스, 아스페르길루스 카르네우스, 아스페르길루스 클라바투스, 아스페르길루스 데플렉투스, 아스페르길루스 피셔리아누스, 아스페르길루스 플라부스, 아스페르길루스 글라우쿠스, 아스페르길루스 니둘란스, 아스페르길루스 오크라세우스, 아스페르길루스 파라시티쿠스, 아스페르길루스 페니실로이데스, 아스페르길루스 레스트릭투스, 아스페르길루스 소자에, 아스페르길루스 타마리, 아스페르길루스 테레우스, 아스페르길루스 우스투스, 아스페르길루스 베르시콜로르;
속 페니실리움, 예컨대 페니실리움 아우란티오그리세움, 페니실리움 빌라이아에, 페니실리움 카멤베르티, 페니실리움 칸디둠, 페니실리움 크리소게눔, 페니실리움 클라비포르메, 페니실리움 콤뮨, 페니실리움 크루스토숨, 페니실리움 디기타툼, 페니실리움 엑스판숨, 페니실리움 푸니쿨로숨, 페니실리움 글라브룸, 페니실리움 글라우쿰, 페니실리움 이탈리쿰, 페니실리움 라쿠싸르미엔테이, 페니실리움 마르네페이, 페니실리움 푸르푸로게눔, 페니실리움 로쿠에포르티, 페니실리움 스톨로니페룸, 페니실리움 울라이엔세, 페니실리움 베루코숨, 페니실리움 비리디카툼;
속 클라비셉스, 예컨대 클라비셉스 아프리카나, 클라비셉스 푸시포르미스, 클라비셉스 히르텔라, 클라비셉스 파스팔리, 클라비셉스 푸르푸레아, 클라비셉스클라비셉스 소르기, 클라비셉스 지자니아에.
속 파에실로미세스, 예컨대 파에실로미세스 디바리카투스
로 이루어지는 군으로부터 선택된다.
아스페르길루스 자포니쿠스, 아스페르길루스 니거, 아스페르길루스 오리자에, 아스페르길루스 니둘란스, 아스페르길루스 푸미가투스, 페니실리움 크리소게눔, 파에실로미세스 디바리카투스, 페니실리움 로쿠에포르티로 이루어지는 군으로부터 선택된 재조합 천연 맥각 알칼로이드 생산자 유기체가 보다 더 바람직하다.
본 발명의 추가의 실시양태는 시클로클라빈을 포함하고/하거나 생산하는 재조합 천연 맥각 알칼로이드 생산자 유기체이고, 여기서 재조합 천연 맥각 알칼로이드 생산자 유기체는 종 아스페르길루스 자포니쿠스 또는 파에실로미세스 디바리카투스의 것이 아니거나, 또는 종, 아스페르길루스 푸미가투스, 또는 클라비셉스 푸르푸레아의 것이다.
본 발명의 추가의 실시양태는 페스투클라빈을 포함하고/하거나 생산하는 재조합 천연 맥각 알칼로이드 생산자 유기체이고, 여기서 재조합 천연 맥각 알칼로이드 생산자 유기체는 종 아스페르길루스 자포니쿠스, 아스페르길루스 푸미가투스, 아스페르길루스 플라부스, 아스페르길루스 테레우스, 클라비셉스 히르텔라, 클라비셉스 아프리카나, 페니실리움 크리소게눔, 및 파에실로미세스 디바리카투스의 것이 아니거나, 또는
종 아스페르길루스 테레우스, 발란시아 안드로포고니스, 발란시아 사이페리, 발란시아 오브텍타, 발란시아 필루아에포르미스, 클라비셉스 푸르푸레아, 클라비셉스 히르텔라, 클라비셉스 푸시포르미스, 클라비셉스 파스팔리, 클라비셉스 지자니아에, 스파셀리아 에리오클로아에, 스파셀리아 텍센시스, 스파셀리아 로벨레시이, 네오티포디움 종 Lp1, 네오티포디움 코에노피알룸, 네오티포디움 롤리이, 페니실리움 시트리눔, 페니실리움 코릴로필룸, 페니실리움 펠루타눔 및 페리글란둘라 이포모에아에의 것이거나, 또는 클라비셉스 푸르푸레아, 클라비셉스 푸시포르미스, 네오티포디움 롤리이 및 페리글란둘라 이포모에아에로 이루어진다.
본 발명의 추가의 실시양태는 아그로클라빈을 포함하고/하거나 생산하는 재조합 천연 맥각 알칼로이드 생산자 유기체이고, 여기서 재조합 천연 맥각 알칼로이드 생산자 유기체는 종 아스페르길루스 테레우스, 발란시아 안드로포고니스, 발란시아 사이페리, 발란시아 오브텍타, 발란시아 필루아에포르미스, 클라비셉스 푸르푸레아, 클라비셉스 히르텔라, 클라비셉스 푸시포르미스, 클라비셉스 파스팔리, 클라비셉스 지자니아에, 스파셀리아 에리오클로아에, 스파셀리아 텍센시스, 스파셀리아 로벨레시이, 네오티포디움 종 Lp1, 네오티포디움 코에노피알룸, 네오티포디움 롤리이, 페니실리움 시트리눔, 페니실리움 코릴로필룸, 페니실리움 펠루타눔 및 페리글란둘라 이포모에아에의 것이 아니거나, 또는 클라비셉스 푸르푸레아, 클라비셉스 푸시포르미스, 네오티포디움 롤리이 및 페리글란둘라 이포모에아에로 이루어지거나,
또는 종 아스페르길루스 자포니쿠스, 아스페르길루스 푸미가투스, 아스페르길루스 플라부스, 아스페르길루스 테레우스, 클라비셉스 히르텔라, 클라비셉스 아프리카나, 페니실리움 크리소게눔, 및 파에실로미세스 디바리카투스의 것이다.
바람직하게는 상기 설명한 임의의 하나의 재조합 천연 맥각 알칼로이드 생산자 유기체의 적어도 하나의 상향조절된 또는 하향조절된 DmaW, EasF, EasE, EasC, EasH, EasD, EasA 또는 EasG 활성은 다음 군으로부터 선택된 적어도 하나의 폴리펩티드, 또는 적어도 하나의 폴리펩티드를 코딩하는 적어도 하나의 폴리뉴클레오티드에 의해 제공된다:
a) 서열 1, 2, 3, 4, 5, 6, 7, 8, 20, 31, 41, 53, 64, 75, 86, 및/또는 95에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드.
d) 서열 102, 122 또는 123, 129, 130, 131, 132, 133, 134, 135 및/또는 136에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드 또는
e) 서열 102, 122, 123, 129, 130, 131, 132, 133, 134, 135 및/또는 136에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드 또는
f) 엄격한 조건 하에 서열 102, 122 또는 123, 123, 129, 130, 131, 132, 133, 134, 135 및/또는 136에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 및
g) 아스페르길루스 자포니쿠스 또는 파에실로미세스 디바리카투스로부터 얻을 수 있는, a) 내지 f) 중의 적어도 하나에 따른 폴리펩티드.
표 9 내지 14는 각각의 DmaW, EasF, EasE, EasC, EasA, EasG, EasD 또는 EasH 활성을 갖는 폴리펩티드 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 표에 언급된 공급원 유기체에 의해 포함되는 나열된 DmaW, EasF, EasE, EasC, EasA, EasG, EasD 또는 EasH 활성을 갖는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드의 조합의 예시적인 비-제한적인 예를 나열한다. 표 내의 각각의 X는 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체에 의해 포함되는 1개 카피의 재조합 폴리뉴클레오티드를 나타낸다.
<표 9>
<표 10>
<표 11>
<표 12>
<표 13>
<표 14>
관련 기술 분야의 통상의 기술자는 표 9 내지 14에 나열된 것과 유사한 추가의 실시양태를 생성할 수 있을 것이다.
특정 바람직한 실시양태는 효모, 바람직하게는 에스. 세레비지아에, 및 시클로클라빈의 생산을 위한 재조합 미생물로서의 그의 용도이고, 여기서 효모, 바람직하게는 에스. 세레비지아에는 다음을 포함한다:
a) 서열 8 및/또는 95에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 8 및/또는 95에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 156에 적어도 90%, 바람직하게는 적어도 95% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 더 바람직하게는 서열 8 및/또는 95에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 8에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, EasH 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트,
b) 서열 2, 31, 32, 33, 34, 35, 36, 37, 38, 39 및/또는 40에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 2, 31, 32, 33, 34, 35, 36, 37, 38, 39 및/또는 40에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 148, 149 및 150에 적어도 95%, 바람직하게는 100% 서열 동일성을 갖고 서열 149의 아미노산 위치 18에 티로신을 갖는 아미노산 서열을 포함하는, EasA 리덕타제 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트,
c) 서열 7, 86, 87, 88, 89, 90, 181, 92, 93 및/또는 94에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 7, 86, 87, 88, 89, 90, 181, 92, 93 및/또는 94에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 183에 적어도 90%, 바람직하게는 적어도 95% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 바람직하게는 서열 7, 86, 87, 88, 89, 90, 181, 92, 93 및/또는 94에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 10에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, EasG 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트,
d) 서열 4, 53, 54, 55, 56, 57, 58, 59, 60, 62 및/또는 63에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 4, 53, 54, 55, 56, 57, 58, 59, 60, 62 및/또는 63에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 152에 적어도 80%, 바람직하게는 적어도 81% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 바람직하게는 서열 4, 53, 54, 55, 56, 57, 58, 59, 60, 62 및/또는 63에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 7에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, EasD 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트,
e) 서열 3 또는 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51 및/또는 52에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 3 또는 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51 및/또는 52에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 192에 적어도 85%, 바람직하게는 적어도 87% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 바람직하게는 서열 3 또는 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51 및/또는 52에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 6에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, EasC 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트,
f) 서열 5, 64, 175, 66, 67, 68, 69, 176, 71, 177, 73 및/또는 178에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 5, 64, 175, 66, 67, 68, 69, 176, 71, 177, 73 및/또는 178에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 191에 적어도 70%, 바람직하게는 적어도 73% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 바람직하게는 서열 5, 64, 175, 66, 67, 68, 69, 176, 71, 177, 73 및/또는 178에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 5에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, EasE 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트,
g) 서열 6, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84 및/또는 85에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 6, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84 및/또는 85에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 154에 적어도 75%, 바람직하게는 적어도 79% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 바람직하게는 서열 6, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84 및/또는 85에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 4에 제시된 정렬에 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, EasF 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트,
h) 서열 1, 19, 20, 21, 22, 23, 24, 25, 26, 27 및/또는 28에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 1, 19, 20, 21, 22, 23, 24, 25, 26, 27 및/또는 28에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 190에 적어도 85%, 바람직하게는 적어도 89% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 바람직하게는 서열 1, 19, 20, 21, 22, 23, 24, 25, 26, 27 및/또는 28에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 3에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, DmaW 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트.
바람직하게는, EasE 활성을 제공하는 폴리펩티드는 효모에서 소포체로의 분류 신호로 기능할 수 있는 N-말단 서열을 갖는다. 재조합 미생물이 에스. 세레비지아에인 경우에, EasE 활성을 제공하는 폴리펩티드는 바람직하게는 에스. 세레비지아에에서 소포체로의 분류 신호로 기능할 수 있는 N-말단 서열을 포함할 것이다.
바람직하게는, DmaW, EasF, EasE, EasC, EasD, EasH, EasA 리덕타제 및/또는 EasG 활성을 제공하는 폴리펩티드를 코딩하는 폴리뉴클레오티드는 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는 폴리뉴클레오티드 서열을 갖는다. 이들 효모의 바람직한 변이체는 표 12에 기재된 실시양태이다.
이들 효모의 추가의 변이체는 아그로클라빈, 페스투클라빈을 생산하기 위해 사용될 수 있다. 효모는 바람직하게는 EasH 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 발현 카세트를 포함하지 않을 것이다.
이들 효모의 바람직한 변이체는 표 13 및 14에 기재된 실시양태이다. 이들 효모의 추가의 변이체는 카노클라빈 I을 생산하기 위해 사용될 수 있지만, EasH, EasA, EasG 또는 EasD 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 발현 카세트를 포함하지 않을 것이다.
이들 효모의 추가의 변이체는 EasH, EasA, EasG 및 EasD 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 발현 카세트를 포함하지만, DmaW, EasF, EasE 또는 EasC 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 발현 카세트는 포함하지 않을 것이다. 이들 효모는 카노클라빈 I이 제공될 때 시클로클라빈을 생산하기 위해 사용될 수 있다.
이들 효모의 추가의 변이체는 EasA, EasG 및 EasD 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 발현 카세트를 포함하지만, DmaW, EasF, EasE, EasC 또는 EasH 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 발현 카세트는 포함하지 않을 것이다. 이들 효모는 카노클라빈 I이 제공될 때 페스투클라빈 또는 아그로클라빈을 생산하기 위해 사용될 수 있다.
바람직한 특정 실시양태는 효모, 바람직하게는 에스. 세레비지아에, 및 시클로클라빈의 생산을 위한 재조합 미생물로서의 그의 용도이고, 여기서 효모, 바람직하게는 에스. 세레비지아에는 다음을 포함한다:
a) 서열 8 및/또는 95에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 8 및/또는 95에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 156에 적어도 90%, 바람직하게는 적어도 95% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 더 바람직하게는 서열 8 및/또는 95에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 8에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, EasH 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트,
b) 서열 2, 31, 32, 33, 34, 35, 36, 37, 38, 39 및/또는 40에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 2, 31, 32, 33, 34, 35, 36, 37, 38, 39 및/또는 40에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 148, 149 및 150에 적어도 95%, 바람직하게는 100% 서열 동일성을 갖고 서열 149의 아미노산 위치 18에 티로신을 갖는 아미노산 서열을 포함하는, EasA 리덕타제 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트,
c) 서열 7, 86, 87, 88, 89, 90, 181, 92, 93 및/또는 94에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 7, 86, 87, 88, 89, 90, 181, 92, 93 및/또는 94에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 183에 적어도 90%, 바람직하게는 적어도 95% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 바람직하게는 서열 7, 86, 87, 88, 89, 90, 181, 92, 93 및/또는 94에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 10에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, EasG 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트,
d) 서열 4, 53, 54, 55, 56, 57, 58, 59, 60, 62 및/또는 63에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 4, 53, 54, 55, 56, 57, 58, 59, 60, 62 및/또는 63에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 152에 적어도 80%, 바람직하게는 적어도 81% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 바람직하게는 서열 4, 53, 54, 55, 56, 57, 58, 59, 60, 62 및/또는 63에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 7에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, EasD 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트,
e) 서열 3 또는 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51 및/또는 52에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 3 또는 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51 및/또는 52에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 192에 적어도 85%, 바람직하게는 적어도 87% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 바람직하게는 서열 3 또는 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51 및/또는 52에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 6에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, EasC 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 2개의 발현 카세트,
f) 서열 5, 64, 175, 66, 67, 68, 69, 176, 71, 177, 73 및/또는 178에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 5, 64, 175, 66, 67, 68, 69, 176, 71, 177, 73 및/또는 178에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 191에 적어도 70%, 바람직하게는 적어도 73% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 바람직하게는 서열 5, 64, 175, 66, 67, 68, 69, 176, 71, 177, 73 및/또는 178에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 5에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, EasE 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 2개의 발현 카세트,
g) 서열 6, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84 및/또는 85에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 6, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84 및/또는 85에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 154에 적어도 75%, 바람직하게는 적어도 79% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 바람직하게는 서열 6, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84 및/또는 85에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 4에 제시된 정렬에 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, EasF 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트,
h) 서열 1, 19, 20, 21, 22, 23, 24, 25, 26, 27 및/또는 28에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 1, 19, 20, 21, 22, 23, 24, 25, 26, 27 및/또는 28에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 190에 적어도 85%, 바람직하게는 적어도 89% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 바람직하게는 서열 1, 19, 20, 21, 22, 23, 24, 25, 26, 27 및/또는 28에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 3에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, DmaW 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트.
바람직하게는, 효모는 다음을 포함한다:
a) 서열 28에 적어도 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 28에 적어도 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 190에 적어도 85%, 바람직하게는 적어도 89% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 바람직하게는 서열 28에 적어도 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 3에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, DmaW 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트 및/또는
b) 서열 76에 적어도 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 76에 적어도 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 154에 적어도 75%, 바람직하게는 적어도 79% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 바람직하게는 서열 76에 적어도 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 4에 제시된 정렬에 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, EasF 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트.
이들 효모의 추가의 변이체는 아그로클라빈, 페스투클라빈을 생산하기 위해 사용될 수 있다. 이들 효모는 바람직하게는 EasH 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 발현 카세트를 포함하지 않을 것이다.
이들 효모의 추가의 변이체는 카노클라빈 I을 생산하기 위해 사용될 수 있지만, EasH, EasA, EasG 또는 EasD 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 발현 카세트를 포함하지 않을 것이다.
본원에 개시된 재조합 미생물 또는 재조합 천연 맥각-알칼로이드 생산자 유기체는 관련 기술 분야에서 사용되는 표준 형질전환 및/또는 돌연변이 프로토콜을 통해 생산될 수 있다. 각각의 종에 가장 효율적인 방법은 문헌, 예를 들어 [Turgeon (2010) Molecular and cell biology methods for fungi, p3-9], [Koushki, MM et al., (2011), AFRICAN JOURNAL OF BIOTECHNOLOGY Vol.10 (41): p7939-7948], [Coyle et al. (2010) Appl Environ Microbiol 76:3898-3903], [Current Protocols in Molecular Biology, Chapter 13. Eds Ausubel F.M. et al. Wiley & Sons, U.K.], 및 [Genome Analysis: A Laboratory Manual, Cloning Systems. Volume 3. Edited by Birren B, Green ED, Klapholz S, Myers RM, Riethman H, Roskams J. New York: Cold Spring Harbor Laboratory Press; 1999:297-565]에서 쉽게 이용가능하다.
이들 방법은 적어도 하나의 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 또는 EasG 활성을 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 수반한다. 상기 폴리뉴클레오티드는 관심있는 미생물의 이미 존재하는 발현 카세트에 통합되도록 표적화될 수 있거나 또는 각각의 미생물에서 폴리뉴클레오티드의 전사 및 번역을 가능하게 하는 재조합 발현 카세트에 포함될 수 있다. 발현 카세트는 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 및 EasG 활성 제공 폴리펩티드 중 적어도 하나를 코딩하는 폴리뉴클레오티드에 작동가능하게 연결된 프로모터 및 종결인자를 포함한다. 적어도 하나의 폴리펩티드를 코딩하는 폴리뉴클레오티드는 바람직하게는 각각의 미생물의 코돈 사용 빈도에 적응될 것이다. 특정 미생물에 사용하기 적합한 프로모터, 종결인자 및 코돈 사용 빈도에 대한 정보는 관련 기술 분야의 통상의 기술자에게 알려져 있다.
실시예에 나열된 것에 추가로 효모, 특히 사카로미세스 세레비지아에에 대해 적합한 구성적 프로모터는 예를 들어 trpC, gpdA, tub2 및 Tef1 프로모터이다.
효모, 특히 사카로미세스 세레비지아에에 대해 적합한 유도가능 프로모터는 예를 들어 Gal1, Gal10, Cup1, Pho5, 및 Met25 프로모터이다.
추가의 프로모터 및 종결인자, 및 클로닝 전략은 예를 들어, 문헌 [Shao et al. (2009) Nucleic Acids Research, Vol. 37, No. 2 e16 (10 pages)]에 설명되어 있다.
상기 설명한 발현 카세트는, 형질전환을 위해 사용될 수 있거나 또는 하나의 게놈 유전자좌 내의 또는 유전자좌에 근접한 몇몇 구축물의 표적화된 통합에 의해 생성될 수 있는 보다 큰 폴리뉴클레오티드에 의해 포함될 수 있거나 포함되지 않을 수 있다. 상기 보다 큰 폴리뉴클레오티드는 동일한 활성을 제공하는 하나 이상의 폴리펩티드를 위한 발현 카세트를 포함할 수 있거나 또는 상이한 활성을 제공하는 하나 이상의 폴리펩티드를 위한 발현 카세트를 포함할 수 있거나, 또는 동일한 활성을 제공하는 하나 이상의 폴리펩티드를 위한 발현 카세트를 포함하고 상이한 활성을 제공하는 하나 이상의 폴리펩티드를 위한 발현 카세트를 포함할 수 있다. 별법으로 또는 상기 설명한 방법 및 폴리뉴클레오티드에 추가로, 재조합 미생물은 수 회 형질전환될 수 있거나, 몇몇 상이한 폴리뉴클레오티드로 동시에 형질전환되어 재조합 미생물의 게놈 내에 2개 이상의 유전자좌에서 발현 카세트를 포함할 수 있다.
따라서, 본 발명의 추가의 실시양태는 상기 실시양태의 임의의 하나에서 설명된 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드 및 상기 실시양태의 임의의 하나에서 설명된 폴리펩티드를 코딩하는 폴리뉴클레오티드의 발현을 위한 발현 카세트 및 적어도 하나의 상기 폴리뉴클레오티드 또는 발현 카세트를 포함하는, 발현 및 형질전환 벡터를 포함하는 벡터이다.
상기 임의의 실시양태에서 설명된 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체는 또한 본 발명에 포함되는, 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드, I 및 카노클라빈 중의 적어도 하나의 생산을 위해 사용될 수 있다. 따라서, 추가의 실시양태는
a) 상기 임의의 하나의 실시양태에서 설명한 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체에서 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나의 생산을 허용하는 조건 하에 상기 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체를 배양하는 단계; 및
b) 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및/또는 카노클라빈 I을 상기 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 및/또는 배양 배지로부터 얻는 단계
를 포함하는, 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나의 생산 방법을 포함한다.
본 발명의 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체는 표면-배양, 침수-배양, 고체-상태 배양, 부생 배양, 진탕 플라스크 배양 또는 의도된 목적에 맞는 임의의 다른 방식으로 성장시킬 수 있다. 바람직하게는, 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체는 침수-배양으로 배양된다.
재조합 미생물 및 재조합 천연 맥각 알칼로이드 생산자 유기체에 대한 성장 조건 및 성장 배지는 관련 기술 분야의 통상의 기술자에게 알려져 있고, 예를 들어 비-재조합 미생물 및 천연 맥각 생산자 유기체의 배양을 위해 사용되는 조건이 적용될 수 있다. 상기 프로토콜 및 방법은 예를 들어 문헌 [Bacon CW (1985) Mycologia Vol 77:418-123], [Schulz B, et al. (1993) Mycol Res 97:1447-1450], [Banks, et al. (1974) Journal of General Microbiology Vol 82: p345-361], [Flieger, M. et al. (1997) Folia Microbiologica Vol. 42: p3-30], [Pazoutova, S. et al. (1981) Journal of Natural Products Vol. 44: p225-235]에 개시되어 있다.
천연 맥각 알칼로이드 생산자 유기체의 배양 방법 및 이에 의한 맥각 알칼로이드의 생산을 유도하거나 향상시키는 방법은 예를 들어 문헌 [Keller, U. and Tudzynski, P. (2002) in: Osiewacz, H.D. (Ed.), The Mycota, Vol. X. "Industrial Applications". Springer, Berlin, pp. 157-181], [Haarmann, T., et al., (2005), Phytochemistry Vol. 66, p1312-1320], 또는 [Furuta, T et al. (1982) Agricultural and Biological Chemistry, Vol 46 (7): p1921-1922], [Tudzynski P. et al., (2001) Applied Microbiology and Biotechnology, Vol. 57 (5-6) p593-605], [Kantorova, M., et al., (2002) Journal of Natural Products, Vol. 65 (7): p1039-1040], [Kozlovsky, A. G.; (2010) Applied Biochemistry and Microbiology, Vol. 46 (5): p525-529], [Kozlovsky, A. G.; (1999) Applied Biochemistry and Microbiology Vol. 35 (5): p477-485], [Narayan V. and Rao K.K., (1982) Biotechnology Letters Vol. 4 (3): p193-196], [Rehacek, Z. et al., (1970) Folia Microbiologica Vol 15 (3): p210-213], [Narayan V. and Rao K.K., (1982) Biotechnology Letters Vol. 4 (3): p193-196], [Rehacek, Z. et al., (1971) Folia Microbiologica Vol 15 (3): p35-40], [Janardhanan K. and Husain A. (1990) Indian Journal of Experimental Biology, Vol. 28(11): p 1054-1057], [Hernandesz M. et al., (1992) Letters in Applied Microbiology Vol. 15(4):p 156-159], [Hernandesz M. et al., (1993) Process Biochemistry Vol. 28(1): p23-27], [Sarkisova M. et al. (1991) Antibiotiki I Khimioperapiya Vol 36(12): p8-10], [Samdani G et al., (1998) Asian Journal of Chemistry Vol.10(2): p373-374]에 개시되어 있다.
예를 들어, 클라비셉스 푸르푸레아에 의한 맥각 알칼로이드의 생산은 바람직하게는 높은 삼투값 및 낮은 인산염 수준을 갖는 성장 배지에 트립토판을 전구체 및 유도자로서 첨가함으로써 개선될 수 있다.
또한, 감염된 호밀 이삭에서 성장한 클라비셉스 푸르푸레아, 클라비셉스 푸시포르미스 또는 클라비셉스 파스팔리에 의한 맥각 알칼로이드의 생산은 맥각 알칼로이드 전구체를 첨가함으로써 개선될 수 있다.
맥각 알칼로이드의 생산을 위해 클라비셉스 푸르푸레아를 사용하는 방법에 대한 추가의 지침은 문헌 [Hulvova H. et al. "Parasitic fungus Claviceps as a source for biotechnological production of ergot alkaloids" Biotechnology Advances 31 (2013) 79-89]에서 볼 수 있다.
재조합 미생물 및 재조합 천연 맥각 알칼로이드 생산자 유기체는 예를 들어 배양 배지를 위한 탄소원으로서 글루코스, 수크로스, 꿀, 덱스트린, 전분, 글리세롤, 당밀, 동물 또는 식물성 오일 등을 사용하여 배양될 수 있다. 또한, 콩가루, 맥아, 옥수수 침지액, 폐 목화씨, 고기 추출물, 폴리펩톤, 맥아 추출물, 효모 추출물, 황산암모늄, 질산나트륨, 우레아 등이 질소원으로 사용될 수 있다. 나트륨, 칼륨, 칼슘, 마그네슘, 코발트, 염소, 인산 (인산수소이칼륨 등), 황산 (황산마그네슘 등) 및 필요한 다른 이온을 생산할 수 있는 무기염의 첨가도 효과적이다. 또한, 다양한 비타민, 예컨대 티아민 (티아민 염산염 등), 아미노산, 예컨대 글루타민 (나트륨 글루타메이트 등), 아스파라긴 (DL-아스파라긴 등), 미량 영양소, 예컨대 뉴클레오티드 등, 및 선택 약물, 예컨대 항생제 등이 또한 필요한 만큼 첨가될 수 있다. 또한, 유기 물질 및 무기 물질은 유기체의 성장을 돕고 맥각 알칼로이드의 생산, 특히 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드, 및/또는 카노클라빈 I의 생산을 촉진하기 위해 적절하게 첨가될 수 있다. 배양 배지의 pH는 예를 들어 대략 pH 5.5 내지 pH 8이다. 배양은 호기 조건 하의 고체 배양 방법, 진탕 배양 방법, 통기 교반 배양 방법 또는 심부 호기 배양 방법과 같은 방법을 사용하여 수행할 수 있지만, 심부 호기 배양 방법이 가장 적합하다. 배양에 적절한 온도는 10℃ 내지 40℃이지만, 많은 경우에 성장은 22℃ 내지 30℃에서 발생한다. 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드, 및/또는 카노클라빈 I의 생산은 배양 배지 및 배양 조건, 또는 사용되는 숙주에 따라 상이하지만, 임의의 배양 방법을 사용하여 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및/또는 카노클라빈 I의 생산의 축적은 일반적으로 2 내지 10일 내에 최대치에 도달하고, 상당히 더 긴 시간, 예를 들어 15, 20, 25, 30, 40, 50일 또는 심지어 그 초과의 시간 동안 진행될 수 있다. 배양은 배양액 내의 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및/또는 카노클라빈 I의 생산량이 그의 최고 수준에 도달할 때 중지하고, 표적 물질은 배양액으로부터 단리되고, 배양 물질로부터 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및/또는 카노클라빈 I의 생성물을 단리하기 위해 정제된다. 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및/또는 카노클라빈 I의 생성물을 단리하기 위해 사용되는 표적 물질은 임의의 유용한 물질, 예를 들어 전체 유기체일 수 있는 유기체 자체 또는 단지 유기체 또는 유기체의 임의의 다른 부분에 의해 생산된 포자, 또는 유기체를 배양하기 위해 사용된 배양 배지일 수 있다. 배양 조건은 예를 들어 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드, 및/또는 카노클라빈 I의 하나 이상의 전구체를 제공함으로써 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드, 및/또는 카노클라빈 I의 생산을 향상시키기 위해 조정될 수 있고, 상기 전구체는 바람직하게는 IPP, 트립토판, DMAPP, DMAT, Me-DMAT 및 카노클라빈 I의 군으로부터 선택된다.
따라서, 본 발명의 추가의 실시양태는
a) 상기 임의의 하나의 실시양태에서 설명한 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체에서 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나의 생산을 허용하는 조건 하에 상기 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체를 배양하는 단계;
b) 상기 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체에게 IPP, 트립토판, DMAPP, DMAT, Me-DMAT 또는 카노클라빈 I의 군으로부터 선택되는 적어도 하나의 화합물을 배양 배지를 통해 제공하는 단계, 및
c) 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 및/또는 배양 배지로부터 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나를 얻는 단계
를 포함하는, 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나의 생산 방법이다.
상기 설명한 방법은 바람직하게는 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및 카노클라빈 I의 군으로부터 선택된 적어도 하나의 화합물을 얻기 위해 사용된다. 바람직하게는, 방법은 시클로클라빈을 얻기 위해 사용된다.
그러나, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드, 카노클라빈 I을 얻는 방법 단계는 특히 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체가 피로클라빈, 푸미클라빈 A 또는 C, 또는 에르고타민과 같은 다른 맥각 알칼로이드의 생산을 위해 추출물로서 적어도 하나의 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I을 사용하는 추가의 유전자를 포함하는 경우에, 생략될 수도 있다. 이어서, 상기 화합물을 유기체 또는 배양 배지로부터 얻는 것이 유용할 수 있다. 예를 들어, 재조합 천연 맥각 알칼로이드 생산자 유기체가 아스페르길루스 푸미가투스인 경우에, 푸미가클라빈 B, 푸미가클라빈 A 또는 푸미가클라빈과 같은 맥각 알칼로이드를 생산하고 얻는 것이 가능할 것이다. 또한, 재조합 천연 맥각 알칼로이드 생산자 유기체가 클라비셉스 종, 바람직하게는 클라비셉스 푸르푸레아, 클라비셉스 파스팔리 또는 클라비셉스 아프리카나인 경우에, 엘리모클라빈, 파스팔산, 리세르그산, 에르고펩탐 및 에르고펩틴의 군으로부터 선택된 하나 이상의 화합물을 생산하고 얻는 것이 가능할 것이다. 하나 이상의 맥각 알칼로이드를 생산하고 그 생산을 위한 전구체로서 적어도 하나의 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I을 이용하는 다른 재조합 천연 맥각 생산자 유기체에게 동일한 원리가 적용될 것이다. 바람직하게는, 천연 맥각 알칼로이드 생산자는 각각의 천연 맥각 알칼로이드 생산자 유기체를 생성하기 위해 사용된 동일한 종 및 균주에 비해 적어도 하나의 상기 다른 맥각 알칼로이드를 보다 다량으로, 보다 고속으로 생산할 것이다. 보다 고속 또는 보다 다량은 동일한 조건 하에서 배양될 때 각각의 천연 맥각 알칼로이드 생산자 유기체를 생성하기 위해 사용된 동일한 종 및 균주에 비해 적어도 5%, 10%, 15%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 또는 적어도 100% 더 많거나 더 빠름을 의미한다.
배양 조건은 사용되는 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체에 대해 상이할 수 있다. 효모, 특히 사카로미세스 세레비지아에의 적합한 성장 조건은 실시예에서 제시된다. 그러나, 상기 성장 조건은 추가로 조정될 수 있다. 예를 들어, 성장 온도는 유기체의 성장을 느리게 하거나 빠르게 하기 위해 조정될 수 있다. 따라서, 본 발명은 적어도 하나의 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및 카노클라빈 I의 생산 방법을 포함하고, 여기서 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체는 10℃ 내지 40℃, 바람직하게는 15℃ 내지 35℃, 보다 바람직하게는 18℃ 내지 32℃, 보다 더 바람직하게는 20℃ 내지 30℃의 온도에서 배양되거나 또는 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체는 10℃ 내지 32℃, 13℃ 내지 32℃, 15℃ 내지 32℃, 16℃ 내지 32℃, 17℃ 내지 32℃, 18℃ 내지 32℃, 19℃ 내지 32℃, 20℃ 내지 32℃, 15℃ 내지 31℃, 15℃ 내지 30℃, 15℃ 내지 29℃, 15℃ 내지 28℃, 15℃ 내지 27℃, 15℃ 내지 26℃, 15℃ 내지 25℃의 온도에서 배양되거나, 또는 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체는 먼저 25℃ 내지 40℃, 바람직하게는 25℃ 내지 35℃의 온도에서, 이어서 10℃ 내지 25℃, 바람직하게는 15℃ 내지 25℃의 온도에서 배양되거나, 또는 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체는 먼저 10℃ 내지 25℃, 바람직하게는 15℃ 내지 25℃의 온도에서, 이어서 25℃ 내지 40℃, 바람직하게는 25℃ 내지 35℃의 온도에서 배양된다.
대체로, 성장 조건은 각각의 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체의 선호도에 따라 선택되지만, 그로부터 벗어나는 것이 유리할 수 있다. 예를 들어, 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및/또는 카노클라빈 I의 생산을 위해 15℃ 내지 25℃, 바람직하게는 20℃의 온도에서 에스. 세레비지아에를 성장시키는 것이 유리할 수 있다.
몇몇 실시양태에서, 각각의 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체, 예를 들어 에스. 세레비지아에의 성장 조건은 성장을 늦추도록 선택된다. 이것은 예를 들어 글리세롤, 트리메틸아민 N-옥시드 (TMAO) 또는 프롤린을 성장 배지에, 예를 들어, 글리세롤을 2.5%, 5% 또는 10%의 농도로, TMAO를 250 mM 또는 500 mM의 농도로, 또는 프롤린을 200 mM의 농도로 첨가함으로써 달성할 수 있다.
추가의 바람직한 실시양태는 높은 세포 밀도를 생성하고/하거나 발효를 연장하기 위해 사용될 수 있는 유가식 배양 조건을 사용한다.
본원에서 설명되는 방법에 의해 생산된 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및/또는 카노클라빈 I은 바람직하게는 추출을 통해 얻지만, 원칙적으로 다른 방법을 통해서도 얻을 수 있다.
한 실시양태에서, 추출은 에틸 아세테이트 (EA), tert-부틸 메틸에테르 (TBME), 클로로포름 (CHCl3) 및 디-클로로메탄 (DCM)의 군으로부터 선택된 화합물을 사용한 추출을 통해 수행한다.
바람직하게는, 추출 방법은 pH=10에서 에틸 아세테이트를 사용한 액체/액체 추출, 보다 바람직하게는 pH=10에서 에틸 아세테이트를 사용한 액체/액체 추출을 포함하고, 여기서 pH는 NaOH로 조정된다.
그러나, 적어도 하나의 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및/또는 카노클라빈 I을, 바람직하게는 pH=3의 HLB 수지, 바람직하게는 pH=3의 디아이온 수지, 바람직하게는 pH=3의 앰벌라이트 1180 수지, 바람직하게는 pH=10의 앰벌라이트 1180 수지, 바람직하게는 pH=3의 앰벌라이트 16N 수지 또는 바람직하게는 pH=10의 앰벌라이트 16N 수지의 군으로부터 선택되는 실리카계 수지 물질을 사용한 고상 추출을 사용한 추출을 포함하는 추출 방법을 통해 얻는 것도 가능하다.
시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및/또는 카노클라빈 I, 또는 다른 클라빈-유형 알칼로이드 또는 다른 맥각 알칼로이드를 얻기 위한 적합한 방법은 관련 기술 분야의 통상의 기술자에게 알려져 있다. 예를 들어, 시클로클라빈을 얻는 방법은 문헌 [Furuta et al., (1982), Agricultural and Biological Chemistry Vol. 46 (7): p1921-1922] 또는 [Stauffacher D. et al. (1969) Tetrahedron Vol.25: p5879-5887]에 개시되어 있다.
맥각 알칼로이드의 분석 방법은 예를 들어, 문헌 [Flieger, M. et al., (1997) Folia Microbiolgica, Vol. 42(1) p 3-29] 및 [Halada, P. et al., (1998) Chemicke Listy Vol.92(7) p538-547]에 설명되어 있다.
본 발명은 적어도 하나의 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데하이드 및 카노클라빈 I의 생산을 위한 또는 상기 설명한 임의의 하나의 방법에서 상기 임의의 하나의 실시양태에서 설명된 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체, 재조합 폴리뉴클레오티드, 벡터의 용도를 포함한다.
본 발명의 추가의 실시양태는 상기 설명한 임의의 하나의 방법에 의해 생산된 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및/또는 카노클라빈 I, 및 본 발명의 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체의 배양에 의해 생산된, 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및 카노클라빈 I의 군으로부터 선택된 적어도 하나의 화합물을 포함하는 배양 배지를 포함한다.
바람직하게는, 배양 배지는 시클로클라빈을 포함하지만, 적어도 하나의 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및 카노클라빈 I은 포함하지 않거나 비교적 낮은 함량으로 포함한다. 동등하게 바람직하게는, 배양 배지는 페스투클라빈을 포함하지만, 적어도 하나의 시클로클라빈, 아그로클라빈, 카노클라빈 알데히드 및 카노클라빈 I은 포함하지 않거나 비교적 낮은 함량으로 포함한다. 동등하게 바람직하게는, 배양 배지는 아그로클라빈을 포함하지만, 적어도 하나의 시클로클라빈, 페스투클라빈, 카노클라빈 알데히드 및 카노클라빈 I은 포함하지 않거나 비교적 낮은 함량으로 포함한다. 또한 동등하게 바람직하게는, 배양 배지는 카노클라빈 알데히드를 포함하지만, 적어도 하나의 시클로클라빈, 페스투클라빈, 아그로클라빈 및 카노클라빈 I은 포함하지 않거나 비교적 낮은 함량으로 포함한다. 또한 동등하게 바람직하게는, 배양 배지는 카노클라빈 I을 포함하지만, 적어도 하나의 시클로클라빈, 페스투클라빈, 아그로클라빈 및 카노클라빈 알데히드는 포함하지 않거나 비교적 낮은 함량으로 포함한다.
비교적 낮은 함량은 문제의 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체의 각각의 대조군 유기체를 배양함으로써 생산된 배양 배지 내의 함량과 비교할 때 적어도 10%, 20%, 30%, 40% 50%, 60%, 70%, 80%, 100% 더 적은 각각의 화합물의 함량을 의미한다.
본 발명은 상기 설명한 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체를 생성하기 위해 사용될 수 있는, 파에실로미세스 디바리카투스로부터 단리된 폴리뉴클레오티드 및 그의 서열 변이체를 추가로 포함한다.
따라서, 본 발명의 한 실시양태는 다음으로 이루어지는 군으로부터 선택되는 하나 이상의 핵산 서열을 포함하는 폴리뉴클레오티드이다:
a) 서열 129, 130, 131, 132, 133, 134, 135 또는 136에 제시된 뉴클레오티드 서열을 갖는 핵산 서열;
b) 서열 20, 31, 41, 53, 64, 75, 86, 95 또는 180에 제시된 아미노산 서열을 갖는 폴리펩티드를 코딩하는 핵산 서열;
c) 서열 129, 130, 131, 132, 133, 134, 135 또는 136에 제시된 핵산 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 동일하고 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 리덕타제, EasA 이소머라제 또는 EasG 활성을 갖는 폴리펩티드를 코딩하는 핵산 서열;
d) DmaW, EasF, EasE, EasC, EasD, EasH, EasA 리덕타제, EasA 이소머라제 또는 EasG 활성을 갖고 서열 20, 31, 41, 53, 64, 75, 86, 95 또는 180에 제시된 아미노산 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 동일한 아미노산 서열을 갖는 폴리펩티드를 코딩하는 핵산 서열; 및
e) 엄격한 조건 하에 a) 내지 d) 중 임의의 하나에 혼성화할 수 있고 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 리덕타제, EasA 이소머라제 또는 EasG 활성을 갖는 폴리펩티드를 코딩하는 핵산 서열.
본 발명의 추가의 실시양태는 상기 설명한 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체를 생성하기 위해 사용될 수 있는 폴리펩티드 및 그의 서열 변이체를 포함한다.
따라서, 본 발명의 한 실시양태는 다음으로 이루어지는 군으로부터 선택된 하나 이상의 핵산 서열을 포함하는 폴리뉴클레오티드이다:
a) 서열 175, 176, 177 또는 178에 제시된 아미노산 서열을 갖는 폴리펩티드를 코딩하는 핵산 서열;
b) EasE 활성을 갖고 서열 175, 176, 177 또는 178에 제시된 아미노산 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 동일한 아미노산 서열을 갖는 폴리펩티드를 코딩하는 핵산 서열;
c) 서열 181에 제시된 아미노산 서열을 갖는 폴리펩티드를 코딩하는 핵산 서열, 및
d) DmaW 활성을 갖고 서열 181에 제시된 아미노산 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 동일한 아미노산 서열을 갖는 폴리펩티드를 코딩하는 핵산 서열.
파에실로미세스 디바리카투스로부터 단리된 상기 폴리뉴클레오티드 및 그의 서열 변이체 및 EasE 또는 DmaW 활성을 갖고 서열 175, 176, 177, 178, 또는 181에 제시된 아미노산 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 동일한 아미노산 서열을 갖는 폴리펩티드를 코딩하는 핵산 서열은 본원에서 설명되는 임의의 하나의 실시양태에서 설명되는 임의의 하나의 폴리뉴클레오티드와, 바람직하게는 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 리덕타제, EasA 이소머라제 또는 EasG 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드와 조합될 수 있다.
폴리뉴클레오티드는 코딩되는 폴리펩티드의 전사 및 번역을 위한 발현 카세트를 생성하기 위해 프로모터 및 종결인자 서열과 추가로 작동가능하게 연결될 수 있다. 또한, 상기 폴리뉴클레오티드 및 발현 카세트는 본 발명의 재조합 미생물 또는 형질전환 및/또는 발현 벡터 내로 도입되고, 예를 들어 재조합 천연 맥각 알칼로이드 생산자 유기체를 생성하기 위해 또는 추가의 정제를 위해, 예를 들어 상기 폴리펩티드를 인식하는 항체를 생산하거나 또는 상기 폴리펩티드의 단백질 결정을 생산하기 위해, 또는 시험관내 시험 및 생산 방법을 위해 폴리펩티드를 사용하기 위해 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 리덕타제, EasA 이소머라제 또는 EasG 활성을 갖는 코딩되는 폴리펩티드를 생산하기 위해 사용될 수 있다. 따라서, 본 발명은 상기 폴리뉴클레오티드를 포함하는 원핵 및 진핵 숙주 세포를 포함한다. 바람직하게는, 상기 숙주 세포는 박테리아 세포, 진균 세포, 또는 효모 세포이다. 본 발명의 추가의 실시양태는 a) DmaW, EasF, EasE, EasC, EasD, EasH, EasA 리덕타제, EasA 이소머라제 또는 EasG 활성을 갖고 서열 20, 31, 41, 53, 64, 75, 86 또는 95에 제시된 아미노산 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 동일한 아미노산 서열을 갖는 폴리펩티드를 코딩하는 핵산 서열에 대한 발현 카세트를 포함하는 숙주 세포를 배양하거나 또는 폴리펩티드의 생산을 허용하는 조건 하에 파에실로미세스 디바리카투스 세포를 배양하는 단계; 및 b) 단계 a)의 숙주 세포 또는 파에실로미세스 디바리카투스 세포로부터 폴리펩티드를 얻는 단계를 포함하는 폴리펩티드의 제조 방법이다.
또한, a) 파에실로미세스 디바리카투스 세포를 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I의 생산을 허용하는 조건 하에 배양하는 단계; 및 b) 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나를 파에실로미세스 디바리카투스 세포 및/또는 배양 배지로부터 얻는 단계를 포함하는, 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I, 또는 이들의 조합물의 제조를 위한 방법이 본 발명에 포함된다.
본 발명의 다른 부분은 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및 카노클라빈 I의 군으로부터 선택된 적어도 하나의 화합물의 제조를 위한 또는 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및 카노클라빈 I의 군으로부터 선택된 적어도 하나의 화합물을 전구체로서 사용함으로써 생산되는 적어도 하나의 맥각 알칼로이드의 제조를 위한,
a) 서열 129, 130, 131, 132, 133, 134, 135 또는 136에 제시된 뉴클레오티드 서열을 갖는 핵산 서열;
b) 서열 20, 31, 41, 53, 64, 75, 86 또는 95에 제시된 아미노산 서열을 갖는 폴리펩티드를 코딩하는 핵산 서열;
c) 서열 129, 130, 131, 132, 133, 134, 135 또는 136에 제시된 핵산 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 동일하고 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 리덕타제, EasA 이소머라제 또는 EasG 활성을 갖는 폴리펩티드를 코딩하는 핵산 서열;
d) DmaW, EasF, EasE, EasC, EasD, EasH, EasA 리덕타제, EasA 이소머라제 또는 EasG 활성을 갖고 서열 20, 31, 41, 53, 64, 75, 86 또는 95에 제시된 아미노산 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 동일한 아미노산 서열을 갖는 폴리펩티드를 코딩하는 핵산 서열; 및
e) 엄격한 조건 하에 a) 내지 d) 중 임의의 하나에 혼성화할 수 있고 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 리덕타제, EasA 이소머라제 또는 EasG 활성을 갖는 폴리펩티드를 코딩하는 핵산 서열
로 이루어지는 군으로부터 선택되는 하나 이상의 핵산 서열을 포함하는 폴리뉴클레오티드의 용도, 또는 상기 폴리뉴클레오티드를 포함하는 벡터, 또는 숙주 세포 또는 파에실로미세스 디바리카투스 세포의 용도를 포함한다.
본 발명의 다른 부분은 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및 카노클라빈 I의 군으로부터 선택된 적어도 하나의 화합물의 제조를 위한 또는 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및 카노클라빈 I의 군으로부터 선택된 적어도 하나의 화합물을 전구체로서 사용하여 생산되는 적어도 하나의 맥각 알칼로이드의 제조를 위한,
a) 서열 175, 176, 177 또는 178에 제시된 아미노산 서열을 갖는 폴리펩티드를 코딩하는 핵산 서열;
b) EasE 활성을 갖고, 서열 175, 176, 177 또는 178에 제시된 아미노산 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 동일한 아미노산 서열을 갖는 폴리펩티드를 코딩하는 핵산 서열;
c) 서열 181에 제시된 아미노산 서열을 갖는 폴리펩티드를 코딩하는 핵산 서열, 및
d) DmaW 활성을 갖고 서열 181에 제시된 아미노산 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 동일한 아미노산 서열을 갖는 폴리펩티드를 코딩하는 핵산 서열
로 이루어지는 군으로부터 선택된 하나 이상의 핵산 서열을 포함하는 폴리뉴클레오티드의 용도, 또는 상기 폴리뉴클레오티드를 포함하는 벡터, 또는 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체, 바람직하게는 아스페르길루스 자포니쿠스, 아스페르길루스 푸미가투스, 파에실로미세스 디바리카투스, 클라비셉스 푸르푸레아, 클라비셉스 파스팔리, 클라비셉스 아프리카누스(Claviceps africanus) 또는 효모 세포의 용도를 포함한다.
본 발명의 추가의 실시양태는 다음으로 이루어지는 군으로부터 선택된 적어도 하나의 폴리펩티드를 발현하는 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체이다:
a) 서열 175, 176, 177 또는 178에 제시된 아미노산 서열을 갖는 폴리펩티드;
b) EasE 활성을 갖고 서열 175, 176, 177 또는 178에 제시된 아미노산 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 동일한 아미노산 서열을 갖는 폴리펩티드;
c) 서열 181에 제시된 아미노산 서열을 갖는 폴리펩티드, 및
d) DmaW 활성을 갖고 서열 181에 제시된 아미노산 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 동일한 아미노산 서열을 갖는 폴리펩티드.
바람직하게는, 상기 폴리펩티드를 발현하는 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체는 아스페르길루스 자포니쿠스, 아스페르길루스 푸미가투스, 파에실로미세스 디바리카투스, 클라비셉스 푸르푸레아, 클라비셉스 파스팔리, 클라비셉스 아프리카누스 또는 효모 세포이다.
실시예
실시예 1: 빵 효모 (사카로미세스 세레비지아에)에서 시클로클라빈의 생산.
물질 및 방법:
본 실험에서 사용한 기본 효모 균주 EYS1456은 다음 유전자형을 가졌다:
CEN.PK 111-61A (MATα MAL2-8C SUC2 his3Δ1 leu2-3_112 ura3-52).
실시예 1에서 사용한 유전자를 표 15에 나열한다:
<표 15>
유전자의 클로닝 :
효모 내에서 발현을 위해 코돈 최적화된 합성 유전자를 제조하였다. 유전자는 아미노산 서열 (서열 102-108) + 번역 중지 코돈을 코딩한다. 합성 동안, 모든 유전자에는 5'-단부에 HindIII 제한 인식 부위를 포함하는 DNA 서열 AAGCTTAAA, 및 3'-단부에 SacII 제한 부위를 포함하는 DNA 서열 CCGCGG이 제공되었다. 유전자를 HindIII 및 SacII를 사용하여, HindIII 및 SacII를 포함하는 다중의 클로닝 부위에 의해 분리된 천연 효모 프로모터 및 종결인자를 함유하는 효모 발현 카세트 (서열 110 및 111) 내로 클로닝하였다. 모든 카세트에는 AscI 제한 부위가 측면에 접해있었다. 유전자 발현 카세트 (서열 110 및 111)는 발현 카세트의 클로닝을 위한 AscI 부위를 제공하기 위해 링커 (서열 113)가 그 내부에 삽입된 pUC18 기반 플라스미드 벡터 (서열 112) 상에 포함되었다.
유전자 발현 카세트를 1 또는 2개의 카세트를 지정된 통합 벡터 (서열 114-118) 내로 서브-클로닝하기 위해 사용하였고, 이것은 또한 효모 선택가능 마커 유전자를 함유하였다. 2개의 발현 카세트의 경우에, 클로닝의 배향은 헤드 투 헤드 (head to head)였고, 이것은 프로모터가 반대 방향으로 전사를 허용할 것임을 의미한다. 마커 및 발현 카세트(들)은 숙주의 게놈 DNA에 상동성을 갖는 서열 (서열 114-118)이 측면에 접해있었다. 상동성 서열이 측면에 접하는, 발현 카세트(들) 및 선택 마커를 포함하는 전체 통합 구축물을, 통합 벡터를 NotI 및 SbfI 제한 효소로 소화시킴으로써 pUC18 기반 백본으로부터 방출시키고, 효모의 형질전환을 위해 사용하였다. 표준 리튬 아세테이트 형질전환 프로토콜을 사용하였다 (Current Protocols; Chapter 13). 정확한 통합을 PCR에 의해 확인하고, 문헌 [Sauer, 1987]에 설명된 바와 같이 Cre 재조합효소를 발현하는 플라스미드로 세포를 형질전환시킨 후에 선택 마커를 제거하였다 (절제하였다). 통합을 위한 표적 부위는 문헌 [Flagfeldt, 2009]에 설명되어 있다. 통합 벡터 및 그들의 통합 구축물의 서열 번호를 표 16a 및 표 16b에 나열한다.
<표 16a>
<표 16b>
경로 어셈블리 :
완전 시클로클라빈 경로, 또는 유전자 EasH가 결여된 경로를 EYS1456 내로 통합함으로써 2개의 효모 균주를 제조하였다. EYS1849는 pEVE2294, pEVE2312, pEVE2343, 및 pEVE2344로부터 구축물을 통합함으로써 제조한 한편 (표 16a 및 16b), EYS1851은 pEVE2294, pEVE2312, pEVE2342, 및 pEVE2344로부터 구축물을 통합함으로써 제조하였다 (표 16a 및 16b). 따라서, 생성되는 균주는 다음 유전자에 대한 발현 카세트를 함유하였다:
EYS1849: AjDmaW_altC, AfEasF, AjEasE, AjEasC, AjEasD, AjEasA, AjEasG, 및 AjEasH.
EYS1851: AjDmaW_altC, AfEasF, AjEasE, AjEasC, AjEasD, AjEasA, 및 AjEasG.
성장 조건 :
조작된 효모 균주 EYS1849 및 EYS1851을 2% 글루코스를 함유하는 표준 SC 브로쓰 (broth) (포미디엄 (ForMedium, 영국 헌스탄톤)) 내에서 성장시켰다. 배양액을 25 ml 배지를 담은 250 ml 진탕 플라스크 내에서 20℃에서 72시간 동안 연속적으로 진탕하면서 성장시켰다.
분석 절차 :
샘플 제조: 효모 배양액을 10분 동안 1000 x g에서 원심분리하였다. 펠렛 및 상청액을 분리하였다. 추가로 정제하지 않고, 5 ㎕의 상청액을 micrOTOF-Q II (브루커 달토닉 게엠베하 (Bruker Daltonik GmbH), 독일 브레멘)에 결합된 UPLC-TOF (워터스 액퀴티(Waters Acquity)™ 초고성능 LC, 워터스 (Waters), 미국 매사추세츠주 밀포드) 내에 주입하였다. 정지상: 컬럼은 액퀴티 UPLC® 브릿지드 (Bridged) 에틸 하이브리드 (BEH) C18 1.7 ㎛ 2.1x100 mm이었다. 액체 크로마토그래피 방법: 이동상 A: H2O + 0.1% 포름산. 이동상 B: 아세토니트릴 + 0.1% 포름산. 진행 조건:
<표 17>
PDA 파라미터: λ 범위: 210 nm 내지 500 nm. 분리능: 1.2 nm. 샘플링 속도: 5 지점/sec. ELSD 파라미터: 게인 (Gain): 50, 기체압: 40 psi, 네뷸라이저 (Nebulizer) 방식: 가열, 파워 (Power) 수준: 80%, 드리프트 (Drift) 튜브: 80℃. TOF 파라미터: 전원: 엔드 플레이트 오프셋(End Plate Offset): -500 V. 모세관: -4500 V. 네뷸라이저: 1.6 bar. 건조 기체: 8.0 L/min. 건조 온도: 180℃. 스캔 (Scan) 방식: MS 스캔. 질량 범위: 80 내지 1000 m/z.
예비 절차 :
샘플 제조: 효모 배양액을 10분 동안 1000 x g에서 원심분리하였다. 펠렛 및 상청액을 분리하였다. 상청액을 10 M NaOH로 pH=10으로 조정하고, 동일 부피의 에틸 아세테이트를 사용하는 액체/액체 추출에 의해 추출하였다. 조 추출물을 진공 하에 건조시키고, 디메틸 술폭시드 (DMSO)로 재구성하여 100 mg/mL의 농도를 얻었다. 이어서, 예비 HPLC 시스템 (워터스, 미국 매사추세츠주 밀포드) 상에서 정제하였다. 정지상: 컬럼은 XBridge™ 예비 C18, 5 ㎛, 19x250 mm 컬럼이었다.
액체 크로마토그래피 방법: 이동상 A: H2O + 0.1% 트리플루오로아세트산. 이동상 B: 아세토니트릴 + 0.1% 트리플루오로아세트산. 진행 조건:
<표 18>
ESI 파라미터: 전원 전압: 모세관: 3.00 kV. 콘(Cone): 30 V. 추출기: 3 V. RF 렌즈: 0.1 V.
전원 온도: 150℃. 탈용매화 온도: 350℃.
결과 :
EYS1849, EYS1851, 및 EYS1456을 상기 설명한 바와 같이 20℃에서 성장시켰다. 상청액을 LC-MS에 의해 분석하였고, EYS1849의 예상된 질량의 시클로클라빈 (m/z = 239.1543+/-0.01 Da)의 추출된 이온 크로마토그램은 5.5 min(+/-0.2)의 체류 시간을 갖는 피크를 보여주었고, 이것은 균주 EYS1851 (또는 EYS1456, 데이타를 제시하지 않음)에서 보이지 않았다. 또 다른 눈에 띄는 피크는 5.91 min의 체류 시간에서 m/z=241.1699+/-0.01 Da의 질량을 갖는 EYS1849 및 EYS1851 내에 존재하였다 (도 11 참조).
체류 시간 5.49 min에서 피크를 상기 설명한 바와 같이 정제하고, LC-MS 및 NMR 분광법에 의해 분석하고, 구조를 해석하였고, 이것은 화합물의 정체가 시클로클라빈임을 확인하였다.
EYS1849의 상청액 내에서 5.49 min에서 용리된 정제된 화합물의 NMR 데이타:
실시예
2: 에이.
푸미가투스
EasF
, 및 에이.
자포니쿠스
DmaW
_
altC
,
EasE
및 EasC를 사용한 빵 효모 (사카로미세스 세레비지아에) 내에서 카노클라빈 I의 생산
.
물질 및 방법 :
본 실험에서 사용한 기본 효모 균주 EYS1456은 다음 유전자형을 가졌다:
CEN.PK 111-61A (MATα MAL2-8C SUC2 his3Δ1 leu2-3_112 ura3-52).
유전자 서열 :
실시예 2에서 사용한 유전자를 표 19에 나열한다.
<표 19>
유전자의 클로닝 :
효모 내에서 발현을 위해 코돈 최적화된 합성 유전자를 제조하였다. 유전자는 아미노산 서열 (서열 102, 107, 106 및 104) + 번역 중지 코돈을 코딩한다. 합성 동안, 모든 유전자에는 5'-단부에 HindIII 제한 인식 부위를 포함하는 DNA 서열 AAGCTTAAA, 및 3'-단부에 SacII 제한 부위를 포함하는 DNA 서열 CCGCGG이 제공되었다. 유전자를 HindIII 및 SacII를 사용하여, HindIII 및 SacII를 포함하는 다중의 클로닝 부위에 의해 분리된 천연 효모 프로모터 및 종결인자를 함유하는 효모 발현 카세트 (서열 110 및 111) 내로 클로닝하였다. 모든 카세트에는 AscI 제한 부위가 측면에 접해있었다. 유전자 발현 카세트 (서열 110 및 111)는 발현 카세트의 클로닝을 위한 AscI 부위를 제공하기 위해 링커 (서열 113)가 그 내부에 삽입된 pUC18 기반 플라스미드 벡터 (서열 112) 상에 포함되었다.
AjDmaW_altC (서열 102) 및 AfEasF (서열 107)을 함유하는 유전자 발현 카세트를 통합 벡터 pEVE2294 (서열 114) 내로 서브-클로닝하기 위해 사용하였고, 이것은 또한 효모 선택가능 마커 유전자를 함유하였다 (표 16a 및 16b 참조). 2개의 발현 카세트는 헤드 투 헤드 배향을 가졌고, 이것은 프로모터가 반대 방향으로 전사를 허용할 것임을 의미한다. 마커 및 발현 카세트는 숙주의 게놈 DNA에 상동성을 갖는 서열이 측면에 접해있었다. 게놈 상동성 서열이 측면에 접하는, 발현 카세트 및 선택 마커를 포함하는 전체 통합 구축물을, NotI 및 SbfI 제한 효소로 소화시킴으로써 pUC18 기반 백본으로부터 방출시키고, 효모의 형질전환을 위해 사용하였다. 표준 리튬 아세테이트 형질전환 프로토콜을 사용하였다 (Current Protocols; Chapter 13). 정확한 통합을 PCR에 의해 확인하고, 문헌 [Sauer, 1987]에 설명된 바와 같이 Cre 재조합효소를 발현하는 플라스미드로 세포를 형질전환시킨 후에 선택 마커를 제거하였다 (절제하였다). 통합을 위한 표적 부위는 문헌 [Flagfeldt, 2009]에 설명되어 있다. 통합 구축물 및 서열 번호를 표 16a 및 16b에 나열한다 (실시예 1 참조).
유전자 AjEasE (서열 106) 및 AjEasC (서열 104)를 PA 및 GC 발현 카세트 (각각 서열 110 및 111) 내로 클로닝한 후, 각각 효모 벡터 pRS315 및 pRS313의 백본 내로 삽입하여, 벡터 pCCL254 및 pCCL256을 제공하였다. pRS 벡터 pRS315 및 pRS313은 이들 원래의 벡터의 Pvu II 부위들 사이에 멀티 클로닝 부위 링커 (서열 113)를 삽입하여, 상기 링커의 AscI 부위 내로 발현 카세트의 클로닝을 허용함으로써 이미 변형되었다. pRS 벡터는 문헌 [Sikorski, 1989]에 설명되어 있다.
경로 어셈블리 :
EYS1456 내로, 카노클라빈 I에 대한 생합성 경로를 도입함으로써 효모 균주를 제조하였다. 먼저, pEVE2294로부터 구축물 (서열 114)을 EYS1456 내로 통합시킴으로써 중간체 균주를 제조하였다 (실시예 1에서 표 16a 및 16b 참조). 생성되는 균주를 빈 벡터 pRS315 및 pRS313으로 추가로 형질전환시켜 CEY2871을 제공하거나, 벡터 pCCL254 및 pCCL256으로 추가로 형질전환시켜 CEY3631을 제공하였다. 따라서, 생성되는 2개의 균주는 다음 유전자에 대한 발현 카세트를 함유하였다:
CEY2871: AjDmaW_altC, 및 AfEasF.
CEY3631: AjDmaW_altC, AfEasF, AjEasE, 및 AjEasC.
성장 조건 :
조작된 효모 균주 CEY2871 및 CEY3631을 2% 글루코스를 함유하고 Leu (류신) 및 His (히스티딘)이 없는 표준 SC 브로쓰 (포미디엄, 영국 헌스탄톤) 내에서 성장시켰다. 배양액을 25 ml 배지를 담은 250 ml 진탕 플라스크 내에서 30℃에서 72시간 동안 연속적으로 진탕하면서 성장시켰다.
분석 절차 :
샘플 제조: 효모 배양액을 10분 동안 1000 x g에서 원심분리하였다. 펠렛 및 상청액을 분리하였다. 추가로 정제하지 않고, 5 ㎕의 상청액을 micrOTOF-Q II (브루커 달토닉 게엠베하, 독일 브레멘)에 결합된 UPLC-TOF (워터스 액퀴티™ 초고성능 LC, 워터스, 미국 매사추세츠주 밀포드) 내에 주입하였다. 정지상: 컬럼은 액퀴티 UPLC® 브릿지드 에틸 하이브리드 (BEH) C18 1.7 ㎛ 2.1x100 mm이었다. 액체 크로마토그래피 방법: 이동상 A: H2O + 0.1% 포름산. 이동상 B: 아세토니트릴 + 0.1% 포름산. 진행 조건:
<표 20>
PDA 파라미터: λ 범위: 210 nm 내지 500 nm. 분리능: 1.2 nm. 샘플링 속도: 5 지점/sec. ELSD 파라미터: 게인: 50, 기체압: 40 psi, 네뷸라이저 방식: 가열, 파워 수준: 80%, 드리프트 튜브: 80℃. TOF 파라미터: 전원: 엔드 플레이트 오프셋: -500 V. 모세관: -4500 V. 네뷸라이저: 1.6 bar. 건조 기체: 8.0 L/min. 건조 온도: 180℃. 스캔 방식: MS 스캔. 질량 범위: 80 내지 1000 m/z.
예비 절차 :
샘플 제조: 효모 배양액을 10분 동안 1000 x g에서 원심분리하였다. 펠렛 및 상청액을 분리하였다. 상청액을 10 M NaOH로 pH=10으로 조정하고, 동일 부피의 에틸 아세테이트를 사용하는 액체/액체 추출에 의해 추출하였다. 조 추출물을 진공 하에 건조시키고, 디메틸 술폭시드 (DMSO)로 재구성하여 100 mg/mL의 농도를 얻었다. 이어서, 예비 HPLC 시스템 (워터스, 미국 매사추세츠주 밀포드) 상에서 정제하였다. 정지상: 컬럼은 XBridge™ 예비 C18, 5 ㎛, 19x250 mm 컬럼이었다.
액체 크로마토그래피 방법: 이동상 A: H2O + 0.1% 트리플루오로아세트산. 이동상 B: 아세토니트릴 + 0.1% 트리플루오로아세트산. 진행 조건:
<표 21>
ESI 파라미터: 전원 전압: 모세관: 3.00 kV. 콘: 30 V. 추출기: 3 V. RF 렌즈: 0.1 V.
전원 온도: 150℃. 탈용매화 온도: 350℃.
결과 :
CEY2871 및 CEY3631의 상청액을 LC-MS에 의해 분석하였다. 예상된 질량의 카노클라빈 I (m/z = 257.164+/-0.01 Da)의 추출된 이온 크로마토그램은 4.4 min(+/-0.2)의 체류 시간을 갖는 균주 CEY3631에서 피크를 보여주었고, 이것은 균주 CEY2871에서 보이지 않았다 (도 12 참조).
체류 시간 4.4 min에서 피크를 상기 설명한 바와 같이 정제하고, LC-MS 및 NMR 분광법에 의해 분석하였다. 상응하는 화합물의 구조를 해석하였고, 이것은 화합물의 정체가 카노클라빈임을 확인하였다.
CEY3631로부터 4.4 min+/-0.2 min에서 용리된 정제된 화합물의 NMR 데이타:
실시예 3: 에이. 푸미가투스로부터 EasF , 에이. 자포니쿠스로부터 DmaW _ altC 및 EasC , 및 에이. 자포니쿠스로부터 EasE 또는 SUMO- EasE를 사용한 카노클라빈 I의 생산.
물질 및 방법 :
본 실험에서 사용한 기본 효모 균주 EYS1456은 다음 유전자형을 가졌다:
CEN.PK 111-61A (MATα MAL2-8C SUC2 his3Δ1 leu2-3_112 ura3-52).
유전자 서열 :
실시예 3에서 사용한 유전자를 표 22에 나열한다.
<표 22>
유전자의 클로닝 :
효모 내에서 발현을 위해 코돈 최적화된 합성 유전자를 제조하였다. 유전자는 아미노산 서열 (서열 102, 107, 106, 119 및 104) + 번역 중지 코돈을 코딩한다. 합성 동안, 모든 유전자에는 5'-단부에 HindIII 제한 인식 부위를 포함하는 DNA 서열 AAGCTTAAA, 및 3'-단부에 SacII 제한 부위를 포함하는 DNA 서열 CCGCGG이 제공되었다. 유전자를 HindIII 및 SacII를 사용하여, HindIII 및 SacII를 포함하는 다중의 클로닝 부위에 의해 분리된 천연 효모 프로모터 및 종결인자를 함유하는 효모 발현 카세트 (서열 110 및 111) 내로 클로닝하였다. 모든 카세트에는 AscI 제한 부위가 측면에 접해있었다. 유전자 발현 카세트 (서열 110 및 111)는 발현 카세트의 클로닝을 위한 AscI 부위를 제공하기 위해 링커 (서열 113)가 그 내부에 삽입된 pUC18 기반 플라스미드 벡터 (서열 112) 상에 포함되었다.
AjDmaW_altC (서열 102) 및 AfEasF (서열 107)을 함유하는 유전자 발현 카세트를 통합 벡터 pEVE2294 (서열 114) 내로 서브-클로닝하기 위해 사용하였고, 이것은 또한 효모 선택가능 마커 유전자를 함유하였다 (실시예 1에서 표 16a 및 16b 참조). 2개의 발현 카세트는 헤드 투 헤드 배향을 가졌고, 이것은 프로모터가 반대 방향으로 전사를 허용할 것임을 의미한다. 마커 및 발현 카세트는 숙주의 게놈 DNA에 상동성을 갖는 서열이 측면에 접해있었다. 게놈 상동성 서열이 측면에 접하는, 발현 카세트 및 선택 마커를 포함하는 전체 통합 구축물을, NotI 및 SbfI 제한 효소로 소화시킴으로써 pUC18 기반 백본으로부터 방출시키고, 효모의 형질전환을 위해 사용하였다. 표준 리튬 아세테이트 형질전환 프로토콜을 사용하였다 (Current Protocols; Chapter 13). 정확한 통합을 PCR에 의해 확인하고, 문헌 [Sauer, 1987]에 설명된 바와 같이 Cre 재조합효소를 발현하는 플라스미드로 세포를 형질전환시킨 후에 선택 마커를 제거하였다 (절제하였다). 통합을 위한 표적 부위는 문헌 [Flagfeldt, 2009]에 설명되어 있다. 통합 구축물 및 서열 번호를 표 16a 및 16b에 나열한다 (실시예 1 참조).
유전자 AjEasE (서열 106) 및 AjEasC (서열 104)를 PA 및 GC 발현 카세트 (각각 서열 110 및 111) 내로 클로닝한 후, 각각 효모 벡터 pRS315 및 pRS313의 백본 내로 삽입하여 벡터 pCCL254 및 pCCL256을 제공하였다. 벡터 pCCL254의 특유한 HindIII 부위 내로, SUMO-태그를 함유하는 합성 DNA 서열을 삽입하여 EasE 효소의 N-말단 SUMO화 버전 (서열 119)을 생성함으로써 벡터 pCCL255를 생성하였다. SUMO-태그의 사용은 예를 들어 문헌 [Peroutka, 2008]에 설명되어 있다.
pRS 벡터 pRS315 및 pRS313은 이들 원래의 벡터의 Pvu II 부위들 사이에 멀티 클로닝 부위 링커 (서열 113)를 삽입하여, 상기 링커의 AscI 부위 내로 발현 카세트의 클로닝을 허용함으로써 이미 변형되었다. pRS 벡터는 문헌 [Sikorski, 1989]에 설명되어 있다.
경로 어셈블리 :
EYS1456 내로, 카노클라빈 I에 대한 생합성 경로를 도입함으로써 효모 균주를 제조하였다. pEVE2294로부터 구축물 (서열 114)을 EYS1456 내로 통합함으로써 중간체 균주를 제조하였다. 생성되는 균주를 벡터 pCCL254 및 pCCL256으로 추가로 형질전환시켜 CEY3631을 제공하거나, 벡터 pCCL254 및 pCCL255로 추가로 형질전환시켜 CEY3645를 제공하였다. 따라서, 생성되는 2개의 균주는 다음 유전자에 대한 발현 카세트를 함유하였다:
CEY3631: AjDmaW_altC, AfEasF, AjEasE, 및 AjEasC.
CEY3645: AjDmaW_altC, AfEasF, SUMO-AjEasE, 및 AjEasC.
성장 조건 :
조작된 효모 균주 CEY3631 및 CEY3645를 2% 글루코스를 함유하고 Leu 및 His이 없는 표준 SC 브로쓰 (포미디엄, 영국 헌스탄톤) 내에서 성장시켰다. 배양액을 25 ml 배지를 담은 250 ml 진탕 플라스크 내에서 30℃에서 72시간 동안 연속적으로 진탕하면서 성장시켰다.
분석 절차 :
샘플 제조: 효모 배양액을 10분 동안 1000 x g에서 원심분리하였다. 펠렛 및 상청액을 분리하였다. 추가로 정제하지 않고, 5 ㎕의 상청액을 micrOTOF-Q II (브루커 달토닉 게엠베하, 독일 브레멘)에 결합된 UPLC-TOF (워터스 액퀴티™ 초고성능 LC, 워터스, 미국 매사추세츠주 밀포드) 내에 주입하였다. 정지상: 컬럼은 액퀴티 UPLC® 브릿지드 에틸 하이브리드 (BEH) C18 1.7 ㎛ 2.1x100 mm이었다. 액체 크로마토그래피 방법: 이동상 A: H2O + 0.1% 포름산. 이동상 B: 아세토니트릴 + 0.1% 포름산. 진행 조건:
<표 23>
PDA 파라미터: λ 범위: 210 nm 내지 500 nm. 분리능: 1.2 nm. 샘플링 속도: 5 지점/sec. ELSD 파라미터: 게인: 50, 기체압: 40 psi, 네뷸라이저 방식: 가열, 파워 수준: 80%, 드리프트 튜브: 80℃. TOF 파라미터: 전원: 엔드 플레이트 오프셋: -500 V. 모세관: -4500 V. 네뷸라이저: 1.6 bar. 건조 기체: 8.0 L/min. 건조 온도: 180℃. 스캔 방식: MS 스캔. 질량 범위: 80 내지 1000 m/z.
결과 :
CEY3631 및 CEY3645의 상청액을 LC-MS에 의해 분석하였다. 카노클라빈 I의 질량 (m/z = 257.164+/-0.01 Da)을 TIC (총 이온 크로마토그램)로부터 추출하였다. 피크는 2.0 min(+/-0.1)의 체류 시간을 가지면서 검출되었고, 이것은 앞서 (실시예 2 참조) 해석된 카노클라빈 I에 대응한다 (도 13 참조).
카노클라빈 I에 대응하는 피크 하 면적을 통합하였고, 이것은 비-SUMO화 버전을 갖는 균주 (CEY3631)에 비해 SUMO화 버전의 EasE를 갖는 균주 (CEY3645) 내에서 대략 27% 증가를 보여주었다 (도 13, 삽입도 참조).
실시예 4: 에이. 푸미가투스 또는 에이. 자포니쿠스로부터 EasF와 조합한 AjDmaW_altC를 사용한 Me-DMAT의 생산.
물질 및 방법 :
본 실험에서 사용한 기본 효모 균주 EYS1456은 다음 유전자형을 가졌다:
CEN.PK 111-61A (MATα MAL2-8C SUC2 his3Δ1 leu2-3_112 ura3-52).
실시예 4에서 사용한 유전자를 표 24에 나열한다:
<표 24>
유전자의 클로닝 :
효모 내에서 발현을 위해 코돈 최적화된 합성 유전자를 제조하였다. 유전자는 아미노산 서열 (서열 102, 107 및 120) + 번역 중지 코돈을 코딩한다. 합성 동안, 모든 유전자에는 5'-단부에 HindIII 제한 인식 부위를 포함하는 DNA 서열 AAGCTTAAA, 및 3'-단부에 SacII 제한 부위를 포함하는 DNA 서열 CCGCGG이 제공되었다. 유전자를 HindIII 및 SacII를 사용하여, HindIII 및 SacII를 포함하는 다중의 클로닝 부위에 의해 분리된 천연 효모 Cup1 프로모터 및 Adh1 종결인자를 함유하는 효모 CA 발현 카세트 (서열 121) 내로 클로닝하였다. 모든 카세트에는 AscI 제한 부위가 측면에 접해있었다. 유전자 발현 카세트 (서열 121)는 발현 카세트의 클로닝을 위한 AscI 부위를 제공하기 위해 링커 (서열 113)가 그 내부에 삽입된 pUC18 기반 플라스미드 벡터 (서열 112) 상에 포함되었다.
유전자 AjDmaW_altC (서열 102)를 CA 발현 카세트 (서열 121) 내로 클로닝한 후, 효모 벡터 pRS316의 백본 내로 삽입하여 벡터 pCCL100을 제공하였다. 유전자 AfEasF (서열 107) 및 AjEasF (서열 120)를 CA 발현 카세트 (서열 121) 내로 클로닝한 후, 개별적으로 효모 벡터 pRS315의 백본 내로 삽입하여 각각 벡터 pCCL98 및 pCCL91을 제공하였다. pRS 벡터 pRS316 및 pRS315는 이들 원래의 벡터의 Pvu II 부위들 사이에 멀티 클로닝 부위 링커 (서열 113)를 삽입하여, 상기 링커의 AscI 부위 내로 발현 카세트의 클로닝을 허용함으로써 이미 변형되었다. pRS 벡터는 문헌 [Sikorski, 1989]에 설명되어 있다.
경로 어셈블리 :
EYS1456 내로, Me-DMAT에 대한 생합성 경로를 도입함으로써 효모 균주를 제조하였다. EYS1456을 벡터 pCCL100 및 pCCL98로 형질전환시켜 CEY2557을 제공하거나, 벡터 pCCL100 및 pCCL91로 형질전환시켜 CEY2625를 제공하였다. 따라서, 생성되는 2개의 균주는 다음 유전자에 대한 발현 카세트를 함유하였다:
CEY2557: AjDmaW_altC 및 AjEasF.
CEY2625: AjDmaW_altC 및 AfEasF.
성장 조건 :
조작된 효모 균주 CEY2557 및 CEY2625를 2% 글루코스를 함유하고 Leu (류신) 및 Ura (우라실)이 없는 표준 SC 브로쓰 (포미디엄, 영국 헌스탄톤) 내에서 성장시켰다. 황산구리를 200 μΜ의 최종 농도로 첨가하였다. 배양액을 25 ml 배지를 담은 250 ml 진탕 플라스크 내에서 30℃에서 72시간 동안 연속적으로 진탕하면서 성장시켰다.
분석 절차 :
샘플 제조: 효모 배양액을 10분 동안 1000 x g에서 원심분리하였다. 펠렛 및 상청액을 분리하였다. 추가로 정제하지 않고, 5 ㎕의 상청액을 micrOTOF-Q II (브루커 달토닉 게엠베하, 독일 브레멘)에 결합된 UPLC-TOF (워터스 액퀴티™ 초고성능 LC, 워터스, 미국 매사추세츠주 밀포드) 내에 주입하였다. 정지상: 컬럼은 액퀴티 UPLC® 브릿지드 에틸 하이브리드 (BEH) C18 1.7 ㎛ 2.1x100 mm이었다. 액체 크로마토그래피 방법: 이동상 A: H2O + 0.1% 포름산. 이동상 B: 아세토니트릴 + 0.1% 포름산. 진행 조건:
<표 25>
PDA 파라미터: λ 범위: 210 nm 내지 500 nm. 분리능: 1.2 nm. 샘플링 속도: 5 지점/sec. ELSD 파라미터: 게인: 50, 기체압: 40 psi, 네뷸라이저 방식: 가열, 파워 수준: 80%, 드리프트 튜브: 80℃. TOF 파라미터: 전원: 엔드 플레이트 오프셋: -500 V. 모세관: -4500 V. 네뷸라이저: 1.6 bar. 건조 기체: 8.0 L/min. 건조 온도: 180℃. 스캔 방식: MS 스캔. 질량 범위: 80 내지 1000 m/z.
예비 절차 :
샘플 제조: 효모 배양액을 10분 동안 1000 x g에서 원심분리하였다. 펠렛 및 상청액을 분리하였다. 상청액을 10 M NaOH로 pH=10으로 조정하고, 동일 부피의 에틸 아세테이트를 사용하는 액체/액체 추출에 의해 추출하였다. 조 추출물을 진공 하에 건조시키고, 디메틸 술폭시드 (DMSO)로 재구성하여 100 mg/mL의 농도를 얻었다. 이어서, 예비 HPLC 시스템 (워터스, 미국 매사추세츠주 밀포드) 상에서 정제하였다: 정지상: 컬럼은 XBridge™ 예비 C18, 5 ㎛, 19x250 mm 컬럼이었다.
액체 크로마토그래피 방법: 이동상 A: H2O + 0.1% 트리플루오로아세트산. 이동상 B: 아세토니트릴 + 0.1% 트리플루오로아세트산. 진행 조건:
<표 26>
ESI 파라미터:
전원 전압: 모세관: 3.00 kV. 콘: 30 V. 추출기: 3 V. RF 렌즈: 0.1 V.
전원 온도: 150℃. 탈용매화 온도: 350℃.
결과 :
CEY2557 및 CEY2625의 상청액을 LC-MS에 의해 분석하였다. 예상된 질량의 Me-DMAT (m/z = 287.175+/-0.01 Da)의 추출된 이온 크로마토그램은 체류 시간 7.8+/-0.2min에서 피크를 보여주었다 (도 14 참조). 상기 피크를 상기 설명한 바와 같이 정제하고, LC-MS 및 NMR 분광법에 의해 분석하고, 구조를 해석하였고, 이것은 화합물의 정체가 Me-DMAT임을 확인하였다.
Me-DMAT에 대응하는 피크 하 면적을 통합하였고, 이것은 에이. 자포니쿠스 버전 (CEY2557)을 갖는 균주에 비해 에이. 푸미가투스 버전의 EasF (CEY2625)을 갖는 균주 (도 15 참조) 내에서 2배 초과의 증가를 보여주었다.
Me-DMAT; CEY2557의 상청액 내에서 5.49 min에서 용리된 정제된 화합물의 NMR 데이타:
실시예 5: CpDmaW , AjDmaW _ altC , 또는 AjDmaW _ trcC와 함께 에이. 푸미가투스로부터 EasF를 사용한 Me- DMAT의 생산.
물질 및 방법 :
본 실험에서 사용한 기본 효모 균주 EYS1456은 다음 유전자형을 가졌다:
CEN.PK 111-61A (MATα MAL2-8C SUC2 his3Δ1 leu2-3_112 ura3-52).
실시예 5에서 사용한 유전자를 표 27에 나열한다:
<표 27>
유전자의 클로닝 :
효모 내에서 발현을 위해 코돈 최적화된 합성 유전자를 제조하였다. 유전자는 아미노산 서열 (서열 102 내지 107, 및 서열 122 내지 123) + 번역 중지 코돈을 코딩한다. 합성 동안, 모든 유전자에는 5'-단부에 HindIII 제한 인식 부위를 포함하는 DNA 서열 AAGCTTAAA, 및 3'-단부에 SacII 제한 부위를 포함하는 DNA 서열 CCGCGG이 제공되었다. 유전자를 HindIII 및 SacII를 사용하여, HindIII 및 SacII를 포함하는 다중의 클로닝 부위에 의해 분리된 천연 효모 Cup1 프로모터 및 Adh1 종결인자를 함유하는 효모 CA 발현 카세트 (서열 121) 내로 클로닝하였다. 모든 카세트에는 AscI 제한 부위가 측면에 접해있었다. 유전자 발현 카세트 (서열 121)는 발현 카세트의 클로닝을 위한 AscI 부위를 제공하기 위해 링커 (서열 113)가 그 내부에 삽입된 pUC18 기반 플라스미드 벡터 (서열 112) 상에 포함되었다.
유전자 AjDmaW_altC (서열 102), AjDmaW_trcC (서열 122) 및 CpDmaW (서열 123)을 CA 발현 카세트 (서열 121) 내로 클로닝한 후, 효모 벡터 pRS316의 백본 내로 삽입하여 벡터 각각 pCCL100, pCCL99, 및 pCCL95를 제공하였다. 유전자 AfEasF를 CA 발현 카세트 (서열 121) 내로 클로닝한 후, 효모 벡터 pRS315의 백본 내로 삽입하여 벡터 pCCL98을 제공하였다. pRS 벡터 pRS316 및 pRS315는 이들 원래의 벡터의 Pvu II 부위들 사이에 멀티 클로닝 부위 링커 (서열 113)를 삽입하여, 상기 링커의 AscI 부위 내로 발현 카세트의 클로닝을 허용함으로써 이미 변형되었다. pRS 벡터는 문헌 [Sikorski, 1989]에 설명되어 있다.
경로 어셈블리 :
EYS1456 내로, Me-DMAT에 대한 생합성 경로를 도입함으로써 효모 균주를 제조하였다. EYS1456을 벡터 pCCL100 및 pCCL98로 형질전환시켜 CEY2557을 제공하거나, 벡터 pCCL99 및 pCCL98로 형질전환시켜 CEY2563을 제공하거나, 벡터 pCCL95 및 pCCL98로 형질전환시켜 CEY2575를 제공하였다. 따라서, 생성되는 3개의 균주는 다음 유전자에 대한 발현 카세트를 함유하였다:
CEY2557: AjDmaW_altC 및 AfEasF.
CEY2563: AjDmaW_trcC 및 AfEasF.
CEY2575: CpDmaW 및 AfEasF.
성장 조건 :
조작된 효모 균주 CEY2557, CEY2563, 및 CEY2575를 2% 글루코스를 함유하고 Leu (류신) 및 Ura (우라실)이 없는 표준 SC 브로쓰 (포미디엄, 영국 헌스탄톤) 내에서 성장시켰다. 황산구리를 200 μΜ의 최종 농도로 첨가하였다. 배양액을 25 ml 배지를 담은 250 ml 진탕 플라스크 내에서 30℃에서 72시간 동안 연속적으로 진탕하면서 성장시켰다.
분석 절차 :
샘플 제조: 효모 배양액을 10분 동안 1000 x g에서 원심분리하였다. 펠렛 및 상청액을 분리하였다. 추가로 정제하지 않고, 5 ㎕의 상청액을 micrOTOF-Q II (브루커 달토닉 게엠베하, 독일 브레멘)에 결합된 UPLC-TOF (워터스 액퀴티™ 초고성능 LC, 워터스, 미국 매사추세츠주 밀포드) 내에 주입하였다. 정지상: 컬럼은 액퀴티 UPLC® 브릿지드 에틸 하이브리드 (BEH) C18 1.7 ㎛ 2.1x100 mm이었다. 액체 크로마토그래피 방법: 이동상 A: H2O + 0.1% 포름산. 이동상 B: 아세토니트릴 + 0.1% 포름산. 진행 조건:
<표 28>
PDA 파라미터: λ 범위: 210 nm 내지 500 nm. 분리능: 1.2 nm. 샘플링 속도: 5 지점/sec. ELSD 파라미터: 게인: 50, 기체압: 40 psi, 네뷸라이저 방식: 가열, 파워 수준: 80%, 드리프트 튜브: 80℃. TOF 파라미터: 전원: 엔드 플레이트 오프셋: -500 V. 모세관: -4500 V. 네뷸라이저: 1.6 bar. 건조 기체: 8.0 L/min. 건조 온도: 180℃. 스캔 방식: MS 스캔. 질량 범위: 80 내지 1000 m/z.
예비 절차 :
샘플 제조: 효모 배양액을 10분 동안 1000 x g에서 원심분리하였다. 펠렛 및 상청액을 분리하였다. 상청액을 10 M NaOH로 pH=10으로 조정하고, 동일 부피의 에틸 아세테이트를 사용하는 액체/액체 추출에 의해 추출하였다. 조 추출물을 진공 하에 건조시키고, 디메틸 술폭시드 (DMSO)로 재구성하여 100 mg/mL의 농도를 얻었다. 이어서, 예비 HPLC 시스템 (워터스, 미국 매사추세츠주 밀포드) 상에서 정제하였다: 정지상: 컬럼은 XBridge™ 예비 C18, 5 ㎛, 19x250 mm 컬럼이었다.
액체 크로마토그래피 방법: 이동상 A: H2O + 0.1% 트리플루오로아세트산. 이동상 B: 아세토니트릴 + 0.1% 트리플루오로아세트산. 진행 조건:
<표 29>
ESI 파라미터:
전원 전압: 모세관: 3.00 kV. 콘: 30 V. 추출기: 3 V. RF 렌즈: 0.1 V.
전원 온도: 150℃. 탈용매화 온도: 350℃.
결과 :
CEY2557, CEY2563, 및 CEY2575의 상청액을 LC-MS에 의해 분석하였다. 피크의 추출된 이온 크로마토그램은 예상된 질량의 Me-DMAT (m/z = 257.164+/-0.01 Da)를 갖는 화합물을 나타냈다 (도 16 참조). 체류 시간 7.8 min에서 상기 피크를 상기 설명한 바와 같이 정제하고, LC-MS 및 NMR 분광법에 의해 분석하였고, 구조를 해석하였고, 이것은 화합물의 정체가 Me-DMAT임을 확인하였다. Me-DMAT에 대응하는 피크 하 면적을 통합하였고 (도 16, 삽입도), 이것은 3가지 모든 균주에 대해 유사한 수준의 Me-DMAT 생산을 보여준다.
실시예 6: 감소된 파르네실 피로인산염 신타제 활성을 갖는 숙주 균주 내에서 에이. 자포니쿠스로부터 DmaW _ altC 및 에이. 푸미가투스로부터 EasF를 사용한 Me-DMAT의 생산.
물질 및 방법 :
본 실험에서 사용한 기본 효모 균주 EYS1456은 다음 유전자형을 가졌다:
CEN.PK 111-61A (MATα MAL2-8C SUC2 his3Δ1 leu2-3_112 ura3-52).
실시예
6에서 사용한 유전자를 표 30에 나열한다
:
<표 30>
유전자의 클로닝 :
효모 내에서 발현을 위해 코돈 최적화된 합성 유전자를 게네아트 아게 (GeneArt AG, 독일 로젠부르크)에 의해 제조하였다. 유전자는 아미노산 서열 (서열 102 및 107) + 번역 중지 코돈을 코딩한다. 합성 동안, 모든 유전자에는 5'-단부에 HindIII 제한 인식 부위를 포함하는 DNA 서열 AAGCTTAAA, 및 3'-단부에 SacII 제한 부위를 포함하는 DNA 서열 CCGCGG이 제공되었다. AjDmaW (서열 102)를 HindIII 및 SacII를 사용하여, HindIII 및 SacII를 포함하는 다중의 클로닝 부위에 의해 분리된 천연 효모 Pgk1 프로모터 및 Adh2 종결인자를 함유하는 효모 PA 발현 카세트 (서열 110) 내로 클로닝하였다. AfEasF를 HindIII 및 SacII를 사용하여, HindIII 및 SacII를 포함하는 다중의 클로닝 부위에 의해 분리된 천연 효모 Tef1 프로모터 및 Eno2 종결인자를 함유하는 효모 TE 발현 카세트 (서열 124) 내로 클로닝하였다. 두 카세트에는 AscI 제한 부위가 측면에 접해있었다. 유전자 발현 카세트 (서열 110 및 124)는 발현 카세트의 클로닝을 위한 AscI 부위를 제공하기 위해 링커 (서열 113)가 그 내부에 삽입된 pUC18 기반 플라스미드 벡터 (서열 112) 상에 포함되었다.
이어서, 유전자 AjDmaW 및 AfEasF를 벡터 pESC-Ura 상에 기반한 단일 2 μΜ 벡터 (애질런트 테크놀로지스 (쉬바이즈) 아게 (Agilent Technologies (Schweiz) AG, 스위스 바젤)) 내로 클로닝하였다: Pgk1 프로모터 및 AfEasF 코딩 서열을 포함하는 PA 발현 카세트의 Bgl II/SacII 단편을 Bgl II 및 SacII로 소화시킨 pESC-Ura 내로 삽입하였다. 이것은 Adh2 종결인자를 pESC-Ura의 Cyc1 종결인자로 대체시킨다. 생성되는 벡터를 Bgl II 및 Sac I로 소화시키고, 벡터 백본을 TE 발현 카세트로부터 Tef1 프로모터를 포함하는, 유사하게 소화시킨 PCR 산물에 라이게이션시켰다. PCR 산물은 5'-단부에 Bgl II 부위, 및 3'-단부에 3개의 부위 Aar I, Pme I, 및 Sac I을 포함하는 PCR 프라이머를 사용하여 제조하였다. 마지막으로, 생성되는 벡터를 Aar I 및 Pme I로 소화시켰다. Aar I은 Bgl II 소화시킨 서열과 일치성인 오버행을 남기도록 설계되었고, Pme I은 평활 말단을 남겼다. 이어서, AjDmaW_altC를 갖는 TE 발현 카세트를 T4 DNA 중합효소를 사용하여 평활하게 만들어진 SacII로 및 이어서 Bgl II로 소화시켰다. 이어서, Tef 1 프로모터 및 AjDmaW_altC 코딩 서열을 포함하는 상기 단편을 백본에 라이게이션시키고, 바로 위에서 설명한 바와 같이 Aar I 및 Pme I로 소화시켰다. 이것은 Eno2 종결인자를 pESC-Ura의 Adh1 종결인자로 대체시켰다. pEVE2075 (서열 125)로 명명한 최종 벡터는 Tef1 프로모터 및 Adh1 종결인자 사이에 AjDmaW, 및 Pgk1 프로모터 및 Cyc1 종결인자 사이에 AfEasF를 함유한다.
감소된 ERG20 활성을 갖는 효모 숙주의 제조 :
효모 균주 EYS1456을 ERG20 조절 영역의 일부를 결실시키고 이를 ScKex2 유전자의 비교적 약한 구성적 천연 프로모터로 대체함으로써 변형시켰다. 먼저, 선택가능 노우르세오스리신 내성 마커 유전자 NatR에 이어, ScKex2 프로모터를 포함하는 벡터 pEVE2049 (서열 126)를 제조하였다. 이어서, NatR-ScKex2 단편을 2개의 프라이머 서열 127 및 128을 사용하는 PCR에 의해 증폭하였다. 서열 127의 5'-단부는 ERG20 번역 출발 부위에 비해 위치 -768 내지 -724에서 효모 숙주의 게놈 서열과 동일하고 (도 17에서 HR1), 3'-단부는 NatR 선택가능 마커의 상류의 pEVE2049 내의 서열과 동일하다. 서열 128의 5'-단부는 ERG20 코딩 서열의 역 상보체 서열과 동일하고 (도 17에서 HR2), 3'-단부는 ScKex2 프로모터의 3'-단부의 역 상보체 서열과 동일하다. PCR 산물을 사용하여 EYS1456을 형질전환시켜, ERG20 프로모터 영역을 ScKEX2 프로모터로 대체시켰다. NatR 선택가능 마커의 사용에 의해 성공적인 통합을 갖는 클론을 선택하였다. 도 17은 통합 구축물 및 게놈 표적 영역의 개략도를 보여준다. 상기 클론, EYS1456을 플라스미드 pEVE2075로 추가로 형질전환시켜 균주 EYS1538 (천연 ERG20 프로모터 영역을 갖는 대조군 균주) 및 EYS1926를 생성하고, 이것은 ScKex2 프로모터로 대체된 천연 ERG20 프로모터를 가졌다.
경로 어셈블리 :
플라스미드 pEVE2075를 보유하는 2개의 효모 균주, EYS1538 및 EYS1926을 상기 설명한 바와 같이 제조하여, Me-DMAT에 대한 생합성 경로로 생성하였다. 따라서, 생성되는 균주는 둘 모두 다음 유전자에 대한 발현 카세트를 함유하였다:
EYS1538 및 EYS1926: AjDmaW_altC 및 AfEasF.
성장 조건:
조작된 효모 균주 EYS1538 및 EYS1926을 2% 글루코스를 함유하고 Ura (우라실)이 없는 표준 SC 브로쓰 (포미디엄, 영국 헌스탄톤) 내에서 성장시켰다. 배양액을 25 ml 배지를 담은 250 ml 진탕 플라스크 내에서 30℃에서 72시간 동안 연속적으로 진탕하면서 성장시켰다.
분석 절차 :
샘플 제조: 효모 배양액을 10분 동안 1000 x g에서 원심분리하였다. 펠렛 및 상청액을 분리하였다. 추가로 정제하지 않고, 5 ㎕의 상청액을 micrOTOF-Q II (브루커 달토닉 게엠베하, 독일 브레멘)에 결합된 UPLC-TOF (워터스 액퀴티™ 초고성능 LC, 워터스, 미국 매사추세츠주 밀포드) 내에 주입하였다. 정지상: 컬럼은 액퀴티 UPLC® 브릿지드 에틸 하이브리드 (BEH) C18 1.7 ㎛ 2.1x100 mm이었다. 액체 크로마토그래피 방법: 이동상 A: H2O + 0.1% 포름산. 이동상 B: 아세토니트릴 + 0.1% 포름산. 진행 조건:
<표 31>
PDA 파라미터: λ 범위: 210 nm 내지 500 nm. 분리능: 1.2 nm. 샘플링 속도: 5 지점/sec. ELSD 파라미터: 게인: 50, 기체압: 40 psi, 네뷸라이저 방식: 가열, 파워 수준: 80%, 드리프트 튜브: 80℃. TOF 파라미터: 전원: 엔드 플레이트 오프셋: -500 V. 모세관: -4500 V. 네뷸라이저: 1.6 bar. 건조 기체: 8.0 L/min. 건조 온도: 180℃. 스캔 방식: MS 스캔. 질량 범위: 80 내지 1000 m/z.
결과 :
EYS1538 및 EYS1926의 상청액을 LC-MS에 의해 분석하였다. 추출된 이온 크로마토그램은 체류 시간 7.8 min에서 예상된 질량의 Me-DMAT (m/z = 257.164+/-0.01 Da)에 대응하는 피크를 나타냈다. 피크는 앞서 정제된 화합물 (실시예 4 참조)을 참조로 Me-DMAT로서 확인되었다. Me-DMAT에 대응하는 피크 하 면적을 통합하였고, 이것은 EYS1538에 비해 EYS1926 내에서 Me-DMAT의 2배 초과의 증가를 보여주었다 (도 18 참조). 상기 증가는 EYS1926 내에서 프로모터 대체의 결과이었고, 이것은 ERG20 하향 조절 및 따라서 Me-DMAT 합성을 위한 DMAPP의 증가된 이용률을 유발하였다.
실시예 7: 피. 디바리카투스 EasH를 포함한 8-단계 이종 경로를 발현함으로써 빵 효모 (사카로미세스 세레비지아에) 내에서 시클로클라빈의 생산.
물질 및 방법 :
본 실험에서 사용한 기본 효모 균주 EYS1456은 다음 유전자형을 가졌다:
CEN.PK 111-61A (MATα MAL2-8C SUC2 his3Δ1 leu2-3_112 ura3-52).
실시예 7에서 사용한 유전자를 표 32에 나열한다:
<표 32>
유전자의 클로닝 :
효모 내에서 발현을 위해 코돈 최적화된 합성 유전자를 디엔에이2.0 인크. (DNA2.0 Inc., 미국 캘리포니아주 멘로 파크) 또는 게네아트 아게 (독일 로젠부르크)에 의해 제조하였다. 유전자는 아미노산 서열 (서열 102 내지 109 및 157) + 번역 중지 코돈을 코딩한다. 합성 동안, 모든 유전자에는 5'-단부에 HindIII 제한 인식 부위를 포함하는 DNA 서열 AAGCTTAAA, 및 3'-단부에 SacII 제한 부위를 포함하는 DNA 서열 CCGCGG이 제공되었다. 유전자를 HindIII 및 SacII를 사용하여, HindIII 및 SacII를 포함하는 다중의 클로닝 부위에 의해 분리된 천연 효모 프로모터 및 종결인자를 함유하는 효모 발현 카세트 (서열 110 및 111) 내로 클로닝하였다. 모든 카세트에는 AscI 제한 부위가 측면에 접해있었다. 유전자 발현 카세트 (서열 110 및 111)는 발현 카세트의 추가의 클로닝을 위한 AscI 부위를 제공하기 위해 링커 (서열 113)가 그 내부에 삽입된 pUC18 기반 플라스미드 벡터 (서열 112) 상에 포함되었다.
유전자 발현 카세트를 1 또는 2개의 카세트를 지정된 통합 벡터 (서열 114 내지 118) 내로 서브-클로닝하기 위해 사용하였고, 이것은 또한 효모 선택가능 마커 유전자를 함유하였다. 2개의 발현 카세트의 경우에, 클로닝의 배향은 헤드 투 헤드였고, 이것은 프로모터가 반대 방향으로 전사를 허용할 것임을 의미한다. 마커 및 발현 카세트(들)은 숙주의 게놈 DNA에 상동성을 갖는 서열 (서열 114 내지 118)이 측면에 접해있었다. 상동성 서열이 측면에 접하는, 발현 카세트(들) 및 선택 마커를 포함하는 전체 통합 구축물을, 통합 벡터를 SbfI 제한 효소로 소화시킴으로써 pUC18 기반 백본으로부터 방출시키고, 효모의 형질전환을 위해 사용하였다. 표준 리튬 아세테이트 형질전환 프로토콜을 사용하였다 (Current Protocols; Chapter 13). 정확한 통합을 PCR에 의해 확인하고, 문헌 [Sauer, 1987]에 설명된 바와 같이 Cre 재조합효소를 발현하는 플라스미드로 세포를 형질전환시킨 후에 선택 마커를 제거하였다 (절제하였다). 통합을 위한 표적 부위는 문헌 [Flagfeldt, 2009]에 설명되어 있다. 통합 벡터 및 그들의 통합 구축물의 서열 번호를 표 33에 나열한다.
<표 33>
또한, 아스페르길루스 자포니쿠스로부터 AjEasH 유전자 (서열 109) 또는 파에실로미세스 디바리카투스로부터 PdEasH 유전자 (서열 번호 028)를 함유하는 2개의 플라스미드를 구축하고, GPD1 프로모터 및 CYC1 종결인자를 사용하는, 발현 카세트 (서열 111)가 설치된 pRS315 벡터 내로 클로닝하였다. 클로닝은 실시예 2에 설명된 바와 같이 수행하였다.
경로 어셈블리 :
EYS1456 내로, pEVE2294, pEVE2312, pEVE2342, 및 pEVE2344로부터의 구축물을 통합함으로써 효모 균주, EYS1851을 제조하였다 (표 33). EYS1851을 각각 PdEasH 또는 AjEasH를 함유하는 pRS 벡터 (상기 참조)로 형질전환시킴으로써 2개의 균주, EYS2098 및 EYS2099를 제조하였다. 따라서, 생성되는 균주는 다음 유전자에 대한 발현 카세트를 함유하였다:
EYS2098: AjDmaW_altC, AfEasF, AjEasE, AjEasC, AjEasD, AjEasA, AjEasG, PdEasH.
EYS2099: AjDmaW_altC, AfEasF, AjEasE, AjEasC, AjEasD, AjEasA, AjEasG, AjEasH.
성장 조건 :
조작된 효모 균주 EYS2098 및 EYS2099를 2% 글루코스를 함유하고 류신이 없는 표준 SC 브로쓰 (포미디엄, 영국 헌스탄톤) 내에서 성장시켰다. 배양액을 25 ml 배지를 담은 250 ml 진탕 플라스크 내에서 20℃에서 72시간 동안 연속적으로 진탕하면서 성장시켰다.
분석 절차 :
샘플 제조: 효모 배양액을 10분 동안 1000 x g에서 원심분리하였다. 펠렛 및 상청액을 분리하였다. 추가로 정제하지 않고, 5 ㎕의 상청액을 micrOTOF-Q II (브루커 달토닉 게엠베하, 독일 브레멘)에 결합된 UPLC-TOF (워터스 액퀴티™ 초고성능 LC, 워터스, 미국 매사추세츠주 밀포드) 내에 주입하였다. 정지상: 컬럼은 액퀴티 UPLC® 브릿지드 에틸 하이브리드 (BEH) C18 1.7 ㎛ 2.1x100 mm이었다. 액체 크로마토그래피 방법: 이동상 A: H2O + 0.1% 포름산. 이동상 B: 아세토니트릴 + 0.1% 포름산. 진행 조건:
<표 34>
PDA 파라미터: λ 범위: 210 nm 내지 500 nm. 분리능: 1.2 nm. 샘플링 속도: 5 지점/sec. ELSD 파라미터: 게인: 50, 기체압: 40 psi, 네뷸라이저 방식: 가열, 파워 수준: 80%, 드리프트 튜브: 80℃. TOF 파라미터: 전원: 엔드 플레이트 오프셋: -500 V. 모세관: -4500 V. 네뷸라이저: 1.6 bar. 건조 기체: 8.0 L/min. 건조 온도: 180℃. 스캔 방식: MS 스캔. 질량 범위: 80 내지 1000 m/z.
결과 :
EYS2098 및 EYS2099로부터 상청액을 LC-MS에 의해 분석하고, 카노클라빈 I (미소량) 및 시클로클라빈의 생산은 순수한 화합물을 참조하여 입증하였다 (화합물 확인에 대해 실시예 1 및 2 참조). 두 균주 내에서 시클로클라빈의 생산 (도 19 참조)은 상기 화합물의 생산에 기여하는 피. 디바리카투스 또는 에이. 자포니쿠스 모두로부터 EasH 유전자의 능력을 보여주고, 이것은 모 균주 EYS1851에 의해 시클로클라빈이 생산되지 않기 때문이다 (실시예 1 참조).
실시예 8: 증가된 이소펜테닐 이인산염:디메틸 -알릴 이인산염 이소머라제 활성 ( Idi1 )을 갖는 숙주 균주 내에서, 에이. 자포니쿠스로부터 DmaW _ altC 및 에이. 푸미가투스로부터 EasF를 사용한 Me-DMAT의 증가된 생산.
물질 및 방법 :
본 실험에서 사용한 기본 효모 균주 EYS1456은 다음 유전자형을 가졌다:
CEN.PK 111-61A (MATα MAL2-8C SUC2 his3Δ1 leu2-3_112 ura3-52).
실시예 8에서 사용한 유전자를 표 35에 나열한다:
<표 35>
유전자의 클로닝 :
번역 중지 코돈을 포함하는, AjDmaW_altC (서열 102) 및 AfEasF (서열 107)의 아미노산 서열을 코딩하는, 효모 내에서 발현을 위해 코돈 최적화된 2개의 합성 유전자를 게네아트 아게 (독일 로젠부르크)에 의해 제조하였다. ScIdi1 (이소펜테닐 이인산염:디메틸알릴 이인산염 이소머라제) (서열 158)를 코딩하는 유전자를 주형으로서 에스. 세레비지아에로부터 게놈 DNA를 사용하는 PCR에 의해 증폭하였다. 합성 (AjDmaW_altC 및 AfEasF) 또는 PCR 증폭 (ScIdi1) 동안, 모든 유전자에는 5'-단부에 HindIII 제한 인식 부위를 포함하는 DNA 서열 AAGCTTAAA, 및 3'-단부에 SacII 제한 부위를 포함하는 DNA 서열 CCGCGG이 제공되었다.
유전자를 HindIII 및 SacII를 사용하여, HindIII 및 SacII를 포함하는 다중의 클로닝 부위에 의해 분리된 천연 효모 프로모터 (각각 Gpd1, Tef1, 및 Tef2) 및 종결인자 (각각 Cyc1, Eno2, 및 Pgi1)를 함유하는 효모 발현 카세트 (서열 110, 124 및 159) 내로 클로닝하였다. 모든 카세트에는 AscI 제한 부위가 측면에 접해있었다. 유전자 발현 카세트는 발현 카세트의 클로닝을 위한 AscI 부위를 제공하기 위해 링커 (서열 113)가 그 내부에 삽입된 pUC18 기반 플라스미드 벡터 (서열 112) 상에 포함되었다. 마지막으로, 유전자를 함유하는 발현 카세트를 AscI 단편으로서 각각 효모 벡터 pRS316, pRS315 및 pRS313의 백본 내로 클로닝하였다. pRS 벡터 pRS316, pRS315 및 pRS313은 이들 원래의 벡터의 Pvu II 부위들 사이에 멀티 클로닝 부위 링커 (서열 113)를 삽입하여, 상기 링커의 AscI 부위 내로 발현 카세트의 클로닝을 허용함으로써 이미 변형되었다. pRS 벡터는 문헌 [Sikorski, 1989]에 설명되어 있다.
경로 어셈블리 :
EYS1456 내로, 유전자 AjDmaW_altC 및 AfEasF를 포함하는 상기 설명한 pRS 벡터를 사용하여 Me-DMAT에 대한 생합성 경로를 도입함으로써 효모 균주, EYS2055를 제조하였다. 상기 균주를 ScIdi1 유전자를 포함하는 제3 pRS 벡터로 추가로 형질전환시켜, 균주 EYS2056을 생성하였다. 따라서, 생성되는 2개의 균주는 다음 유전자에 대한 발현 카세트를 함유하였다:
EYS2055: AjDmaW_altC, 및 AfEasF.
EYS2056: AjDmaW_altC, AfEasF, 및 ScIdi1.
성장 조건 :
조작된 효모 균주 EYS2055 및 EYS2056을 2% 글루코스를 함유하고 His (히스티딘), Leu (류신) 및 Ura (우라실)이 없는 표준 SC 브로쓰 (포미디엄, 영국 헌스탄톤) 내에서 성장시켰다. 배양액을 25 ml 배지를 담은 250 ml 진탕 플라스크 내에서 20℃ 또는 30℃에서 72시간 동안 연속적으로 진탕하면서 성장시켰다.
분석 절차 :
샘플 제조: 효모 배양액을 10분 동안 1000 x g에서 원심분리하였다. 펠렛 및 상청액을 분리하였다. 추가로 정제하지 않고, 5 ㎕의 상청액을 micrOTOF-Q II (브루커 달토닉 게엠베하, 독일 브레멘)에 결합된 UPLC-TOF (워터스 액퀴티™ 초고성능 LC, 워터스, 미국 매사추세츠주 밀포드) 내에 주입하였다. 정지상: 컬럼은 액퀴티 UPLC® 브릿지드 에틸 하이브리드 (BEH) C18 1.7 ㎛ 2.1x100 mm이었다. 액체 크로마토그래피 방법: 이동상 A: H2O + 0.1% 포름산. 이동상 B: 아세토니트릴 + 0.1% 포름산. 진행 조건:
<표 36>
PDA 파라미터: λ 범위: 210 nm 내지 500 nm. 분리능: 1.2 nm. 샘플링 속도: 5 지점/sec. ELSD 파라미터: 게인: 50, 기체압: 40 psi, 네뷸라이저 방식: 가열, 파워 수준: 80%, 드리프트 튜브: 80℃. TOF 파라미터: 전원: 엔드 플레이트 오프셋: -500 V. 모세관: -4500 V. 네뷸라이저: 1.6 bar. 건조 기체: 8.0 L/min. 건조 온도: 180℃. 스캔 방식: MS 스캔. 질량 범위: 80 내지 1000 m/z.
결과 :
EYS2055 및 EYS2056의 상청액을 LC-MS에 의해 분석하였다. 예상된 질량의 DMAT (m/z = 273.159+/-0.01 Da) 및 Me-DMAT (m/z = 287.175+/-0.01 Da)의 이온 크로마토그램을 추출하고, 피크 하 면적을 통합하였다. 결과 (도 20a 및 20b 참조)는 두 온도에서 EYS2055에 비해 균주 EYS2056 내에서 Me-DMAT 생산의 증가를 보여주었다. 증가는 EYS2055의 정상 야생형 발현 수준에 비해 ScIdi1의 과다발현에 대응하였다.
실시예 9: 전체 길이 이종 경로의 추가의 카피의 유전자를 발현함으로써 시클로클라빈의 증가된 생산.
물질 및 방법 :
본 실험에서 사용한 기본 효모 균주 EYS1456은 다음 유전자형을 가졌다:
CEN.PK 111-61A (MATα MAL2-8C SUC2 his3Δ1 leu2-3_112 ura3-52).
실시예 9에서 사용한 유전자를 표 37에 나열한다:
<표 37>
유전자의 클로닝 :
효모 내에서 발현을 위해 코돈 최적화된 합성 유전자를 디엔에이2.0 인크. (미국 캘리포니아주 멘로 파크) 또는 게네아트 아게 (독일 로젠부르크)에 의해 제조하였다. 유전자는 아미노산 서열 (서열 102 내지 109) + 번역 중지 코돈을 코딩한다. 합성 동안, 모든 유전자에는 5'-단부에 HindIII 제한 인식 부위를 포함하는 DNA 서열 AAGCTTAAA, 및 3'-단부에 SacII 제한 부위를 포함하는 DNA 서열 CCGCGG이 제공되었다. 유전자를 HindIII 및 SacII를 사용하여, HindIII 및 SacII를 포함하는 다중의 클로닝 부위에 의해 분리된 천연 효모 프로모터 및 종결인자를 함유하는 효모 발현 카세트 (서열 110 및 111) 내로 클로닝하였다. 모든 카세트에는 AscI 제한 부위가 측면에 접해있었다. 유전자 발현 카세트 (서열 110 및 111)는 발현 카세트의 클로닝을 위한 AscI 부위를 제공하기 위해 링커 (서열 113)가 그 내부에 삽입된 pUC18 기반 플라스미드 벡터 (서열 112) 상에 포함되었다.
유전자 발현 카세트를 1 또는 2개의 카세트를 지정된 통합 벡터 (서열 114, 115, 117, 118 및 160) 내로 서브-클로닝하기 위해 사용하였고, 이것은 또한 효모 선택가능 마커 유전자를 함유하였다. 클로닝의 배향은 헤드 투 헤드였고, 이것은 프로모터가 반대 방향으로 전사를 허용할 것임을 의미한다. 마커 및 발현 카세트(들)은 숙주의 게놈 DNA에 상동성을 갖는 서열이 측면에 접해있었다. 상동성 서열이 측면에 접하는, 발현 카세트(들) 및 선택 마커를 포함하는 최종 통합 구축물 (서열 114, 115, 117, 118 및 160)을, 통합 벡터를 SbfI 제한 효소로 소화시킴으로써 pUC18 기반 백본으로부터 방출시키고, 효모의 형질전환을 위해 사용하였다. 표준 리튬 아세테이트 형질전환 프로토콜을 사용하였다 (Current Protocols; Chapter 13). 정확한 통합을 PCR에 의해 확인하고, 문헌 [Sauer, 1987]에 설명된 바와 같이 Cre 재조합효소를 발현하는 플라스미드로 세포를 형질전환시킨 후에 선택 마커를 제거하였다 (절제하였다). 통합을 위한 표적 부위는 문헌 [Flagfeldt, 2009]에 설명되어 있다. 통합 벡터 및 그들의 통합 구축물의 서열 번호를 표 38에 나열한다.
<표 38>
경로 어셈블리 :
EYS1456 내로, 단일 카피의 완전 시클로클라빈 경로 (EYS1934) 또는 완전 경로 + 추가의 카피의 유전자 AjEasC (EYS2206)를 통합함으로써 2개의 효모 균주를 제조하였다. EYS1934는 pEVE2294, pEVE2312, pEVE2343, 및 pEVE2344로부터 구축물을 통합시킴으로써 제조한 한편 (표 38), EYS2206은 pEVE2294, pEVE2312, pEVE2343, pEVE2344, 및 pEVE2656으로부터 구축물을 통합시킴으로써 제조하였다 (표 38). 따라서, 생성되는 균주는 다음 유전자에 대한 발현 카세트를 함유하였다:
EYS1934: AjDmaW_altC, AfEasF, AjEasE, AjEasC, AjEasD, AjEasA, AjEasG, 및 AjEasH.
EYS2206: AjDmaW_altC, AfEasF, AjEasE, 2 x AjEasC, AjEasD, AjEasA, AjEasG, 및 AjEasH.
성장 조건 :
조작된 효모 균주 EYS1934 및 EYS2206을 2% 글루코스를 함유하는 표준 SC 브로쓰 (포미디엄, 영국 헌스탄톤) 내에서 성장시켰다. 배양액을 25 ml 배지를 담은 250 ml 진탕 플라스크 내에서 30℃에서 72시간 동안 연속적으로 진탕하면서 성장시켰다.
분석 절차 :
샘플 제조: 효모 배양액을 10분 동안 1000 x g에서 원심분리하였다. 펠렛 및 상청액을 분리하였다. 추가로 정제하지 않고, 5 ㎕의 상청액을 micrOTOF-Q II (브루커 달토닉 게엠베하, 독일 브레멘)에 결합된 UPLC-TOF (워터스 액퀴티™ 초고성능 LC, 워터스, 미국 매사추세츠주 밀포드) 내에 주입하였다. 정지상: 컬럼은 액퀴티 UPLC® 브릿지드 에틸 하이브리드 (BEH) C18 1.7 ㎛ 2.1x100 mm이었다. 액체 크로마토그래피 방법: 이동상 A: H2O + 0.1% 포름산. 이동상 B: 아세토니트릴 + 0.1% 포름산. 진행 조건:
<표 39>
PDA 파라미터: λ 범위: 210 nm 내지 500 nm. 분리능: 1.2 nm. 샘플링 속도: 5 지점/sec. ELSD 파라미터: 게인: 50, 기체압: 40 psi, 네뷸라이저 방식: 가열, 파워 수준: 80%, 드리프트 튜브: 80℃. TOF 파라미터: 전원: 엔드 플레이트 오프셋: -500 V. 모세관: -4500 V. 네뷸라이저: 1.6 bar. 건조 기체: 8.0 L/min. 건조 온도: 180℃. 스캔 방식: MS 스캔. 질량 범위: 80 내지 1000 m/z.
결과 :
EYS1934 및 EY2206을 상기 설명한 바와 같이 30℃에서 성장시켰다. 상청액을 LC-MS에 의해 분석하고, 예상된 질량의 시클로클라빈 (m/z = 239.1543+/-0.01 Da)의 이온 크로마토그램을 추출하였다. 또한, 약 241의 m/z (m/z = 241.1699+/-0.01 Da) 후에 MW241로 명명한 경로의 제2 산물의 크로마토그램을 추출하였다. 피크 하 면적을 통합하였고, 결과의 비교는 EYS1934에 비해 EYS2206 내에서 시클로클라빈 (CCL) 및 페스투클라빈 (MW241) 모두의 증가된 생산을 보여주었다. 상기 증가 (도 21 참조)는 EYS1934에 비해 EYS2206 내에서 추가의 카피의 AjEasC의 발현 효과인 것으로 여겨졌다.
실시예 10: 엔. 롤리이 EasE를 포함한 8-단계 이종 경로를 발현함으로써 빵 효모 (사카로미세스 세레비지아에) 내에서 시클로클라빈의 생산.
물질 및 방법 :
본 실험에서 사용한 기본 효모 균주 EYS1456은 다음 유전자형을 가졌다:
CEN.PK 111-61A (MATα MAL2-8C SUC2 his3Δ1 leu2-3_112 ura3-52).
실시예 10에서 사용한 유전자를 표 40에 나열한다:
<표 40>
유전자의 클로닝 :
효모 내에서 발현을 위해 코돈 최적화된 합성 유전자. 유전자는 아미노산 서열 (서열 102 내지 109 및 161) + 번역 중지 코돈을 코딩한다. 합성 동안, 모든 유전자에는 5'-단부에 HindIII 제한 인식 부위를 포함하는 DNA 서열 AAGCTTAAA, 및 3'-단부에 SacII 제한 부위를 포함하는 DNA 서열 CCGCGG이 제공되었다. 유전자 (서열 102, 107, 105, 109, 103, 및 108)를 HindIII 및 SacII를 사용하여, HindIII 및 SacII를 포함하는 다중의 클로닝 부위에 의해 분리된 천연 효모 프로모터 및 종결인자를 함유하는 효모 발현 카세트 (서열 110 및 111) 내로 클로닝하였다. 모든 카세트에는 AscI 제한 부위가 측면에 접해있었다. 유전자 발현 카세트 (서열 110 및 111)는 발현 카세트의 클로닝을 위한 AscI 부위를 제공하기 위해 링커 (서열 113)가 그 내부에 삽입된 pUC18 기반 플라스미드 벡터 (서열 112) 상에 포함되었다.
유전자 발현 카세트를 지정된 통합 벡터 (서열 114, 117, 및 118) 내로 카세트를 쌍으로 서브-클로닝하기 위해 사용하였고, 이것은 또한 효모 선택가능 마커 유전자를 함유하였다. 클로닝의 배향은 헤드 투 헤드였고, 이것은 프로모터가 반대 방향으로 전사를 허용할 것임을 의미한다. 마커 및 발현 카세트(들)은 숙주의 게놈 DNA에 상동성을 갖는 서열이 측면에 접해있었다. 상동성 서열이 측면에 접하는, 발현 카세트(들) 및 선택 마커를 포함하는 전체 통합 구축물 (서열 114, 117, 및 118)을, 통합 벡터를 SbfI 제한 효소로 소화시킴으로써 pUC18 기반 백본으로부터 방출시키고, 효모의 형질전환을 위해 사용하였다. 표준 리튬 아세테이트 형질전환 프로토콜을 사용하였다 (Current Protocols; Chapter 13). 정확한 통합을 PCR에 의해 확인하고, 문헌 [Sauer, 1987]에 설명된 바와 같이 Cre 재조합효소를 발현하는 플라스미드로 세포를 형질전환시킨 후에 선택 마커를 제거하였다 (절제하였다). 통합을 위한 표적 부위는 문헌 [Flagfeldt, 2009]에 설명되어 있다. 통합 벡터 및 그들의 통합 구축물의 서열 번호를 표 41에 나열한다.
<표 41>
또한, 3개의 플라스미드를 구축하였다: 하나는 pRS313 내로 클로닝된 AjEasC 유전자 (서열 104)를 포함하였다. pRS313에는 천연 효모 Cup1 프로모터 및 Adh1 종결인자를 포함하는, C/A 발현 카세트 (서열 121)가 이미 설치되었다. 아스페르길루스 자포니쿠스로부터 AjEasE 유전자 (서열 106) 또는 네오티포디움 롤리이로부터 NIEasE 유전자 (서열 161)를 함유하는 다른 2개의 플라스미드를, C/A 발현 카세트 (서열 121)가 또한 설치된 pRS315 벡터 내로 각각의 유전자를 클로닝함으로써 제조하였다. 클로닝은 실시예 2에 설명된 바와 같이 수행하였다.
경로 어셈블리 :
EYS1456 내로, pEVE2294, pEVE2343, 및 pEVE2344로부터 구축물을 통합시킴으로써 효모 균주, EYS1937을 제조하였다 (표 41). 이어서, EYS1937을, 각각 AjEasE 또는 NIEasE와 함께 AjEasC를 함유하는 pRS 벡터 (상기 참조)로 형질전환시킴으로써 2개의 균주, EYS2124 및 EYS2125를 제조하였다. 따라서, 생성되는 균주는 다음 유전자에 대한 발현 카세트를 함유하였다:
EYS2124: AjDmaW_altC, AfEasF, AjEasE, AjEasC, AjEasD, AjEasA, AjEasG, AjEasH.
EYS2125: AjDmaW_altC, AfEasF, NIEasE, AjEasC, AjEasD, AjEasA, AjEasG, AjEasH.
성장 조건 :
조작된 효모 균주 EYS2124 및 EYS2125를 2% 글루코스를 함유하고 류신 및 히스티딘이 없는 표준 SC 브로쓰 (포미디엄, 영국 헌스탄톤) 내에서 성장시켰다. 황산구리를 200 μΜ의 최종 농도로 첨가하였다. 배양액을 25 ml 배지를 담은 250 ml 진탕 플라스크 내에서 20℃에서 72시간 동안 연속적으로 진탕하면서 성장시켰다.
분석 절차 :
샘플 제조: 효모 배양액을 10분 동안 1000 x g에서 원심분리하였다. 펠렛 및 상청액을 분리하였다. 추가로 정제하지 않고, 5 ㎕의 상청액을 micrOTOF-Q II (브루커 달토닉 게엠베하, 독일 브레멘)에 결합된 UPLC-TOF (워터스 액퀴티™ 초고성능 LC, 워터스, 미국 매사추세츠주 밀포드) 내에 주입하였다. 정지상: 컬럼은 액퀴티 UPLC® 브릿지드 에틸 하이브리드 (BEH) C18 1.7 ㎛ 2.1x100 mm이었다. 액체 크로마토그래피 방법: 이동상 A: H2O + 0.1% 포름산. 이동상 B: 아세토니트릴 + 0.1% 포름산. 진행 조건:
<표 42>
PDA 파라미터: λ 범위: 210 nm 내지 500 nm. 분리능: 1.2 nm. 샘플링 속도: 5 지점/sec. ELSD 파라미터: 게인: 50, 기체압: 40 psi, 네뷸라이저 방식: 가열, 파워 수준: 80%, 드리프트 튜브: 80℃. TOF 파라미터: 전원: 엔드 플레이트 오프셋: -500 V. 모세관: -4500 V. 네뷸라이저: 1.6 bar. 건조 기체: 8.0 L/min. 건조 온도: 180℃. 스캔 방식: MS 스캔. 질량 범위: 80 내지 1000 m/z.
결과 :
EYS2124 및 EYS2125로부터 상청액을 LC-MS에 의해 분석하고, 예상된 질량의 시클로클라빈 (m/z = 239.1543+/-0.01 Da)의 이온 크로마토그램을 추출하였다. 또한, 약 241의 m/z (m/z = 241.1699+/-0.01 Da) 후에 MW241 (페스투클라빈)로 명명한 경로의 제2 산물의 크로마토그램을 추출하였다. 피크 하 면적을 통합하였고, 결과의 비교는 2개의 균주 내에서 시클로클라빈 (CCL) 및 페스투클라빈 모두의 생산을 보여주었고, 여기서 생산 수준은 EYS2125에 비해 EYS2124 내에서 더 높았다 (도 22 참조). 이것은 사용된 조건 하에, 에이. 자포니쿠스로부터 EasE 유전자가 엔. 롤리이로부터 EasE보다 더 효율적이지만, 2개의 EasE 상동체는 모두 기능적이고 CCL 및 MW241의 생산을 일으켰음을 보여준다.
실시예 11: 상이한 코돈 사용 빈도를 갖는 합성 EasE 유전자를 포함한 8-단계 이종 경로를 발현함으로써 빵 효모 ( 사카로미세스 세레비지아에) 내에서 시클로클라 빈의 생산.
물질 및 방법 :
본 실험에서 사용한 기본 효모 균주 EYS1456은 다음 유전자형을 가졌다:
CEN.PK 111-61A (MATα MAL2-8C SUC2 his3Δ1 leu2-3_112 ura3-52).
실시예 11에서 사용한 유전자를 표 43에 나열한다:
<표 43>
유전자의 클로닝 :
효모 내에서 발현을 위해 코돈 최적화된 합성 유전자. 유전자는 아미노산 서열 (서열 102 내지 109, 및 162 내지 165) + 번역 중지 코돈을 코딩한다. 합성 동안, 모든 유전자에는 5'-단부에 HindIII 제한 인식 부위를 포함하는 DNA 서열 AAGCTTAAA, 및 3'-단부에 SacII 제한 부위를 포함하는 DNA 서열 CCGCGG이 제공되었다. 유전자 (서열 102, 107, 105, 109, 103, 및 108)를 HindIII 및 SacII를 사용하여, HindIII 및 SacII를 포함하는 다중의 클로닝 부위에 의해 분리된 천연 효모 프로모터 및 종결인자를 함유하는 효모 발현 카세트 (서열 110 및 111) 내로 클로닝하였다. 모든 카세트에는 AscI 제한 부위가 측면에 접해있었다. 유전자 발현 카세트 (서열 110 및 111)는 발현 카세트의 클로닝을 위한 AscI 부위를 제공하기 위해 링커 (서열 113)가 그 내부에 삽입된 pUC18 기반 플라스미드 벡터 (서열 112) 상에 포함되었다.
유전자 발현 카세트를 지정된 통합 벡터 (서열 114, 117 및 118) 내로 카세트를 쌍으로 서브-클로닝하기 위해 사용하였고, 이것은 또한 효모 선택가능 마커 유전자를 함유하였다. 클로닝의 배향은 헤드 투 헤드였고, 이것은 프로모터가 반대 방향으로 전사를 허용할 것임을 의미한다. 마커 및 발현 카세트(들)은 숙주의 게놈 DNA에 상동성을 갖는 서열이 측면에 접해있었다. 상동성 서열이 측면에 접하는, 발현 카세트(들) 및 선택 마커를 포함하는 전체 통합 구축물 (서열 114, 117 및 118)을, 통합 벡터를 SbfI 제한 효소로 소화시킴으로써 pUC18 기반 백본으로부터 방출시키고, 효모의 형질전환을 위해 사용하였다. 표준 리튬 아세테이트 형질전환 프로토콜을 사용하였다 (Current Protocols; Chapter 13). 정확한 통합을 PCR에 의해 확인하고, 문헌 [Sauer, 1987]에 설명된 바와 같이 Cre 재조합효소를 발현하는 플라스미드로 세포를 형질전환시킨 후에 선택 마커를 제거하였다 (절제하였다). 통합을 위한 표적 부위는 문헌 [Flagfeldt, 2009]에 설명되어 있다. 통합 벡터 및 그들의 통합 구축물의 서열 번호를 표 44에 나열한다.
<표 44>
또한, 6개의 플라스미드를 구축하였다: 하나는 pRS313 내로 클로닝된 AjEasC 유전자 (서열 104)를 포함하였다. pRS313에는 천연 효모 Cup1 프로모터 및 Adh1 종결인자를 포함하는 C/A 발현 카세트 (서열 121)가 이미 설치되었다. 5개의 별도의 코돈 사용 빈도를 갖는 합성된 아스페르길루스 자포니쿠스로부터 AjEasE 유전자의 변이체를 함유하는 (서열 106 및 162 내지 165) 다른 5개는 C/A 발현 카세트 (서열 121)가 또한 설치된 pRS315 벡터 내로 클로닝함으로써 제조하였다. 클로닝은 실시예 2에 설명된 바와 같이 수행하였다.
경로 어셈블리 :
EYS1456 내로, pEVE2294, pEVE2343, 및 pEVE2344로부터 구축물을 통합시킴으로써 효모 균주, EYS1937을 제조하였다 (표 44). 이어서, EYS1937을 각각의 AjEasE 유전자의 1개의 버전과 함께 AjEasC를 함유하는 pRS 벡터 (상기 참조)로 형질전환시킴으로써 5개의 균주, EYS2124, EYS2127, EYS2147, EYS2148 및 EYS2149를 제조하였다. 따라서, 생성되는 균주는 다음 유전자에 대한 발현 카세트를 함유하였다:
EYS2124: AjDmaW_altC, AfEasF, AjEasE (서열 106), AjEasC, AjEasD, AjEasA, AjEasG, AjEasH.
EYS2127: AjDmaW_altC, AfEasF, AjEasE (서열 162), AjEasC, AjEasD, AjEasA, AjEasG, AjEasH.
EYS2147: AjDmaW_altC, AfEasF, AjEasE (서열 163), AjEasC, AjEasD, AjEasA, AjEasG, AjEasH.
EYS2148: AjDmaW_altC, AfEasF, AjEasE (서열 164), AjEasC, AjEasD, AjEasA, AjEasG, AjEasH.
EYS2149: AjDmaW_altC, AfEasF, AjEasE (서열 165), AjEasC, AjEasD, AjEasA, AjEasG, AjEasH.
성장 조건 :
조작된 효모 균주 EYS2124, EYS2127, EYS2147, EYS2148 및 EYS2149를 2% 글루코스를 함유하고 류신 및 히스티딘이 없는 표준 SC 브로쓰 (포미디엄, 영국 헌스탄톤) 내에서 성장시켰다. 황산구리를 200 μΜ의 최종 농도로 첨가하였다. 배양액을 25 ml 배지를 담은 250 ml 진탕 플라스크 내에서 30℃에서 72시간 동안 연속적으로 진탕하면서 성장시켰다.
분석 절차 :
샘플 제조: 효모 배양액을 10분 동안 1000 x g에서 원심분리하였다. 펠렛 및 상청액을 분리하였다. 추가로 정제하지 않고, 5 ㎕의 상청액을 micrOTOF-Q II (브루커 달토닉 게엠베하, 독일 브레멘)에 결합된 UPLC-TOF (워터스 액퀴티™ 초고성능 LC, 워터스, 미국 매사추세츠주 밀포드) 내에 주입하였다. 정지상: 컬럼은 액퀴티 UPLC® 브릿지드 에틸 하이브리드 (BEH) C18 1.7 ㎛ 2.1x100 mm이었다. 액체 크로마토그래피 방법: 이동상 A: H2O + 0.1% 포름산. 이동상 B: 아세토니트릴 + 0.1% 포름산. 진행 조건:
<표 45>
PDA 파라미터: λ 범위: 210 nm 내지 500 nm. 분리능: 1.2 nm. 샘플링 속도: 5 지점/sec. ELSD 파라미터: 게인: 50, 기체압: 40 psi, 네뷸라이저 방식: 가열, 파워 수준: 80%, 드리프트 튜브: 80℃. TOF 파라미터: 전원: 엔드 플레이트 오프셋: -500 V. 모세관: -4500 V. 네뷸라이저: 1.6 bar. 건조 기체: 8.0 L/min. 건조 온도: 180℃. 스캔 방식: MS 스캔. 질량 범위: 80 내지 1000 m/z.
결과 :
EYS2124, EYS2127, EYS2147, EYS2148 및 EYS2149로부터 상청액을 LC-MS에 의해 분석하고, 예상된 질량의 시클로클라빈 (m/z = 239.1543+/-0.01 Da)의 이온 크로마토그램을 추출하였다. 또한, 약 241의 m/z (m/z = 241.1699+/-0.01 Da) 때문에 MW241로 명명한 경로의 제2 산물의 크로마토그램을 추출하였다. 피크 하 면적을 통합하였고, 결과는 5개의 모든 균주 내에서 시클로클라빈 (CCL) 및 페스투클라빈 (MW241) 모두의 다양한 수준의 생산을 보여주었다 (도 23 참조). 시험된 모든 EasE 유전자 버전은 동일한 아미노산 서열을 코딩하고, 따라서, 화합물 생산 수준의 변동은 상기 합성 EasE 유전자에서 사용된 상이한 DNA 코돈 선택을 반영한다.
실시예 12: 빵 효모 ( 사카로미세스 세레비지아에) 내에서 8-단계 이종 경로를 발현하고, 효모를 삼투물질의 존재 하에 성장시킴으로써 시클로클라빈의 증가된 생산.
삼투물질의 존재는 몇몇 경우에 단백질의 정확한 폴딩을 개선하는 것으로 나타났다 (Bandyopadhyay 2012, Burkewitz 2012). 여기서, 본 발명자들은 삼투물질 글리세롤이 성장 배지에 첨가될 때 효모 내에서 이종 발현된 경로에 기반한 시클로클라빈의 생산을 개선시킬 수 있음을 보여준다.
물질 및 방법 :
본 실험에서 사용한 기본 효모 균주 EYS1456은 다음 유전자형을 가졌다:
CEN.PK 111-61A (MATα MAL2-8C SUC2 his3Δ1 leu2-3_112 ura3-52).
실시예 12에서 사용한 유전자를 표 46에 나열한다:
<표 46>
유전자의 클로닝 :
효모 내에서 발현을 위해 코돈 최적화된 합성 유전자를 디엔에이2.0 인크. (미국 캘리포니아주 멘로 파크) 또는 게네아트 아게 (독일 로젠부르크)에 의해 제조하였다. 유전자는 아미노산 서열 (서열 102 내지 109) + 번역 중지 코돈을 코딩한다. 합성 동안, 모든 유전자에는 5'-단부에 HindIII 제한 인식 부위를 포함하는 DNA 서열 AAGCTTAAA, 및 3'-단부에 SacII 제한 부위를 포함하는 DNA 서열 CCGCGG이 제공되었다. 유전자를 HindIII 및 SacII를 사용하여, HindIII 및 SacII를 포함하는 다중의 클로닝 부위에 의해 분리된 천연 효모 프로모터 및 종결인자를 함유하는 효모 발현 카세트 (서열 110 및 111) 내로 클로닝하였다. 모든 카세트에는 AscI 제한 부위가 측면에 접해있었다. 유전자 발현 카세트 (서열 110 및 111)는 발현 카세트의 클로닝을 위한 AscI 부위를 제공하기 위해 링커 (서열 113)가 그 내부에 삽입된 pUC18 기반 플라스미드 벡터 (서열 112) 상에 포함되었다.
유전자 발현 카세트를 2개의 카세트를 각각 지정된 통합 벡터 (서열 114, 115, 117 및 118) 내로 서브-클로닝하기 위해 사용하였고, 이것은 또한 효모 선택가능 마커 유전자를 함유하였다. 클로닝의 배향은 헤드 투 헤드였고, 이것은 프로모터가 반대 방향으로 전사를 허용할 것임을 의미한다. 마커 및 발현 카세트(들)은 숙주의 게놈 DNA에 상동성을 갖는 서열이 측면에 접해있었다. 상동성 서열이 측면에 접하는, 발현 카세트(들) 및 선택 마커를 포함하는 최종 통합 구축물 (서열 114, 115, 117 및 118)을, 통합 벡터를 SbfI 제한 효소로 소화시킴으로써 pUC18 기반 백본으로부터 방출시키고, 효모의 형질전환을 위해 사용하였다. 표준 리튬 아세테이트 형질전환 프로토콜을 사용하였다 (Current Protocols; Chapter 13). 정확한 통합을 PCR에 의해 확인하고, 문헌 [Sauer, 1987]에 설명된 바와 같이 Cre 재조합효소를 발현하는 플라스미드로 세포를 형질전환시킨 후에 선택 마커를 제거하였다 (절제하였다). 통합을 위한 표적 부위는 문헌 [Flagfeldt, 2009]에 설명되어 있다. 통합 벡터 및 그들의 통합 구축물의 서열 번호를 표 47에 나열한다.
<표 47>
경로 어셈블리 :
EYS1456 내로, pEVE2294, pEVE2312, pEVE2343, 및 pEVE2344로부터 구축물을 사용하여 (표 47) 단일 카피의 완전 시클로클라빈 경로를 통합시킴으로써, 및 추가로 3개의 빈 플라스미드 pRS313, pRS315, 및 pRS316 (상기 설명한)을 도입함으로써 효모 균주 (EYS2006)를 제조하였다. 따라서, 생성되는 균주는 다음 유전자에 대한 발현 카세트를 함유하였다:
EYS2006: AjDmaW_altC, AfEasF, AjEasE, AjEasC, AjEasD, AjEasA, AjEasG, 및 AjEasH.
성장 조건 :
조작된 효모 균주 EYS2006을 2% 글루코스를 함유하고 류신, 히스티딘 및 우라실이 없는 표준 SC 브로쓰 (포미디엄, 영국 헌스탄톤) 내에서 성장시켰다. EYS2006의 배양액을 글리세롤 없이 또는 2.5%, 5%, 및 10% 글리세롤과 함께 성장시켰다. 0.8 mL의 배양액을 바이오렉터 (BioLector) 발효기 (엠2피-랩스 (M2P-labs, 독일 바에스바일러)) 내에서 30℃에서 72시간 동안 성장시켰다.
분석 절차 :
샘플 제조: 효모 배양액을 10분 동안 1000 x g에서 원심분리하였다. 펠렛 및 상청액을 분리하였다. 추가로 정제하지 않고, 5 ㎕의 상청액을 micrOTOF-Q II (브루커 달토닉 게엠베하, 독일 브레멘)에 결합된 UPLC-TOF (워터스 액퀴티™ 초고성능 LC, 워터스, 미국 매사추세츠주 밀포드) 내에 주입하였다. 정지상: 컬럼은 액퀴티 UPLC® 브릿지드 에틸 하이브리드 (BEH) C18 1.7 ㎛ 2.1x100 mm이었다. 액체 크로마토그래피 방법: 이동상 A: H2O + 0.1% 포름산. 이동상 B: 아세토니트릴 + 0.1% 포름산. 진행 조건:
<표 48>
PDA 파라미터: λ 범위: 210 nm 내지 500 nm. 분리능: 1.2 nm. 샘플링 속도: 5 지점/sec. ELSD 파라미터: 게인: 50, 기체압: 40 psi, 네뷸라이저 방식: 가열, 파워 수준: 80%, 드리프트 튜브: 80℃. TOF 파라미터: 전원: 엔드 플레이트 오프셋: -500 V. 모세관: -4500 V. 네뷸라이저: 1.6 bar. 건조 기체: 8.0 L/min. 건조 온도: 180℃. 스캔 방식: MS 스캔. 질량 범위: 80 내지 1000 m/z.
결과 :
EYS2006을 상기 설명한 바와 같이 30℃에서 성장시켰다. 상청액을 LC-MS에 의해 분석하고, 예상된 질량의 시클로클라빈 (m/z = 239.1543+/-0.01 Da)의 이온 크로마토그램을 추출하였다. 또한, 약 241의 m/z (m/z = 241.1699+/-0.01 Da) 때문에 페스투클라빈 (MW241)인 경로의 제2 산물의 크로마토그램을 추출하였다. 피크 하 면적을 통합하고 비교하였다 (도 24 참조). 대조군 (글리세롤 없이 성장시킨 EYS2006)에 비교할 때, 글리세롤과 함께 성장시킨 배양액은 보다 많은 시클로클라빈 및 페스투클라빈을 생산하였다. 시험한 조건 하에, 화합물 생산의 수준은 증가하는 농도의 글리세롤과 함께 증가하였고, 여기서 최고 생산 수준은 10% 글리세롤에서 얻어졌다.
실시예 13: 이종 CCL 경로로부터 다중의 카피의 유전자를 발현시키고, 숙주 Pdi1 및 Fad1을 과다발현시킴으로써 시클로클라빈의 증가된 생산.
물질 및 방법 :
본 실험에서 사용한 기본 효모 균주 EYS1456은 다음 유전자형을 가졌다:
CEN.PK 111-61A (MATα MAL2-8C SUC2 his3Δ1 leu2-3_112 ura3-52).
실시예 13에서 사용한 유전자를 표 49에 나열한다:
<표 49>
유전자의 클로닝 :
효모 내에서 발현을 위해 코돈 최적화된 합성 유전자. 유전자는 아미노산 서열 (서열 102 내지 109) + 번역 중지 코돈을 코딩한다. 천연 효모 (에스. 세레비지아에) 유전자 Pdi1 (서열 166) 및 Fad1 (서열 167)을 게놈 DNA로부터 증폭하였다. 합성 동안, 모든 유전자에는 5'-단부에 HindIII 제한 인식 부위를 포함하는 DNA 서열 AAGCTTAAA, 및 3'-단부에 SacII 제한 부위를 포함하는 DNA 서열 CCGCGG이 제공되었다. 유전자를 HindIII 및 SacII를 사용하여, HindIII 및 SacII를 포함하는 다중의 클로닝 부위에 의해 분리된 천연 효모 프로모터 및 종결인자를 함유하는 효모 발현 카세트 (서열 110, 111, 159 및 168) 내로 클로닝하였다. 모든 카세트에는 AscI 제한 부위가 측면에 접해있었다. 유전자 발현 카세트 (서열 110, 111, 159, 및 168)는 발현 카세트의 클로닝을 위한 AscI 부위를 제공하기 위해 링커 (서열 113)가 그 내부에 삽입된 pUC18 기반 플라스미드 벡터 (서열 112) 상에 포함되었다.
유전자 발현 카세트를 1, 2 또는 3개의 카세트를 지정된 통합 벡터 (서열 114, 115, 117, 118, 160, 169, 170, 및 171) 내로 서브-클로닝하기 위해 사용하였고, 이것은 또한 효모 선택가능 마커 유전자를 함유하였다. 마커 및 발현 카세트(들)은 숙주의 게놈 DNA에 상동성을 갖는 서열이 측면에 접해있었다. 상동성 서열이 측면에 접하는, 발현 카세트(들) 및 선택 마커를 포함하는 최종 통합 구축물 (서열 114, 115, 117, 118, 160, 169, 170, 및 171)을, 통합 벡터를 SbfI 제한 효소로 소화시킴으로써 pUC18 기반 백본으로부터 방출시키고, 효모의 형질전환을 위해 사용하였다. 표준 리튬 아세테이트 형질전환 프로토콜을 사용하였다 (Current Protocols; Chapter 13). 정확한 통합을 PCR에 의해 확인하였다. 통합을 위한 표적 부위는 문헌 [Flagfeldt, 2009]에 설명되어 있다. 통합 벡터 및 그들의 통합 구축물의 서열 번호를 표 50a 및 50b에 나열한다.
<표 50a>
<표 50b>
경로 어셈블리 :
EYS1456 내로, 단일 카피의 완전 시클로클라빈 경로 (EYS1934), 또는 완전 경로 + 추가의 카피의 CCL 경로 유전자 및 숙주 유전자를 통합시킴으로써 3개의 효모 균주를 제조하였다. 따라서, EYS1934는 pEVE2294, pEVE2312, pEVE2343, 및 pEVE2344로부터 구축물을 통합시킴으로써 제조하고 (표 50), EYS2209는 pEVE2294, pEVE2312, pEVE2343, pEVE2344, pEVE2656, 및 pEVE2658로부터 구축물을 통합시킴으로써 제조하고 (표 50a 및 50b), EYS2297은 pEVE2294, pEVE2312, pEVE2343, pEVE2344, pEVE2656, pEVE2658, pEVE2682, 및 pEVE2723으로부터 구축물을 통합시킴으로써 제조하였다 (표 50a 및 50b). EYS1934에서는 Cre 재조합효소 (문헌 [Sauer, 1987]에 설명된)를 발현함으로써 선택 마커를 제거하였고 (절제하였고), EYS2209 및 EYS2297의 제조를 위한 중간체로서 역할을 하였다. 따라서, 생성되는 균주는 다음 유전자에 대한 발현 카세트를 함유하였다:
EYS1934: AjDmaW_altC, AfEasF, AjEasE, AjEasC, AjEasD, AjEasA, AjEasG, 및 AjEasH.
EYS2209: AjDmaW_altC, AfEasF, AjEasE, 2 x AjEasC, AjEasD, AjEasA, AjEasG, AjEasH, ScPdi1, 및 ScFad1.
EYS2325: 2 x AjDmaW_altC, AfEasF, 2 x AjEasE, 3 x AjEasC, AjEasD, AjEasA, AjEasG, 3 x AjEasH, ScPdi1, 및 ScFad1.
성장 조건 :
조작된 효모 균주 EYS1934 및 EYS2209를 바이오렉터 (엠2피 랩스, 독일 바에스바일러) 내에서 2% 글루코스를 함유하는 표준 SC+모든 브로쓰 (포미디엄, 영국 헌스탄톤) 내에서 성장시켰다. 성장기를 연장하기 위해, 10% 글리세롤을 성장 배지에 첨가하였다. 배양액을 30℃에서 72시간 동안 연속적으로 진탕하면서 성장시켰다. 후속적으로, EYS2209 및 EYS2297을 동일한 조건에서 성장시켰다.
분석 절차 :
샘플 제조: 효모 배양액을 10분 동안 1000 x g에서 원심분리하였다. 펠렛 및 상청액을 분리하였다. 추가로 정제하지 않고, 5 ㎕의 상청액을 micrOTOF-Q II (브루커 달토닉 게엠베하, 독일 브레멘)에 결합된 UPLC-TOF (워터스 액퀴티™ 초고성능 LC, 워터스, 미국 매사추세츠주 밀포드) 내에 주입하였다. 정지상: 컬럼은 액퀴티 UPLC® 브릿지드 에틸 하이브리드 (BEH) C18 1.7 ㎛ 2.1x100 mm이었다. 액체 크로마토그래피 방법: 이동상 A: H2O + 0.1% 포름산. 이동상 B: 아세토니트릴 + 0.1% 포름산. 진행 조건:
<표 51>
PDA 파라미터: λ 범위: 210 nm 내지 500 nm. 분리능: 1.2 nm. 샘플링 속도: 5 지점/sec. ELSD 파라미터: 게인: 50, 기체압: 40 psi, 네뷸라이저 방식: 가열, 파워 수준: 80%, 드리프트 튜브: 80℃. TOF 파라미터: 전원: 엔드 플레이트 오프셋: -500 V. 모세관: -4500 V. 네뷸라이저: 1.6 bar. 건조 기체: 8.0 L/min. 건조 온도: 180℃. 스캔 방식: MS 스캔. 질량 범위: 80 내지 1000 m/z.
결과 :
EYS1934 및 EYS2209의 배양 상청액을 LC-MS에 의해 분석하고, 예상된 질량의 시클로클라빈 (m/z = 239.1543+/-0.01 Da)의 이온 크로마토그램을 추출하였다. 피크 하 면적을 통합하였고, 결과의 비교는 EYS1934에 비해 EYS2209 내에서 시클로클라빈의 증가된 생산을 보여주었고 (도 25, 좌측), 상기 조건 하에 거의 12 mg/L의 역가에 도달하였다. 상기 증가는 EYS1934에 비해 EYS2209 내에서 추가의 카피의 Aj_EasC, Sc_Pdi1, 및 Sc_Fad1의 발현 효과인 것으로 여겨졌다.
동일한 성장 조건, 및 추출 및 분석 방법을 이용하여, EYS2209 및 EYS2297을 비교하였고, 이것은 EYS2209에 비해 EYS2297 내에서 22 mg/L로 CCL 역가의 추가의 증가를 보여주었다 (도 25, 우측). 상기 증가는 EYS2209에 비해 EYS2297 내에서 추가의 카피의 Aj_DmaW_altC, Aj_EasC, Aj_EasE, 및 Aj_EasH (x2)의 발현 효과인 것으로 여겨졌다.
실시예 14: 글루코스의 연속 공급에 의한 시클로클라빈의 증가된 생산.
물질 및 방법 :
본 실험에서 사용한 기본 효모 균주 EYS2325는 다음 유전자형을 가졌다:
MATα MAL2-8C SUC2 his3Δ1 leu2-3_112 ura3-52 YERCΔ8::AjEasC/KanMX
YHRCΔ14::ScPdi1/ScFad1/BleR YMRWΔ15::AjEasH/AjDmaW/HygR YN-
RCΔ9::AjEasH/AjEasC/AjEasE/Nat YORWΔ17::AjDmaW/AfEasF
YORWΔ22::AjEasH/AjEasD YPRCt3::AjEasG/AjEasA YPRCΔ15::AjEasE/AjEasC
[ARS/CEN/URA3] [ARS/CEN/HIS3] [ARS/CEN/LEU2]
이것은 균주 EYS2297 (실시예 13에 설명된)을 3개의 빈 플라스미드 pRS313, pRS315, 및 pRS316 (Sikorski, 1989)로 형질전환시켜 균주 원영양체를 제조하였다.
균주 EYS2325를 사용한 CCL의 생산을 바이오매스 생산에 결합시키고 (내부 관찰), 따라서 본 발명자들은 높은 바이오매스를 생산하는 것을 목표로 하는 유가식 발효 공정을 설정하였다. 통상적인 공급 방식을 선택하였고, 통기 및 교반 방식은 발효적 대사를 피하고, 글루코스 축적 및 에탄올 및/또는 아세테이트 형성을 최소화하는 것을 목표로 하였다.
공정은 합성 완전 배지 (SC)를 사용하여 배치로서 출발하였고, 그 후에 글루코스, 염, 비타민, 미량 금속 및 아미노산을 함유하는 공급을 개시하였다. 설명된 발효 공정을 위해 사용된 플랫폼은 1 L의 최대 작업 부피를 갖는 용기를 사용하는 인포스 아게 (Infors AG, 스위스 모트밍겐)의 멀티포스(Multifors) 2이었다. 출발 부피는 0.32 L이고, 공급기 동안 미리 정해진 지수적 공급 방식을 이용하여 총 435.6 mL의 공급물을 첨가하여, 대략 755 mL의 발효 브로쓰의 최종 부피를 얻었다.
씨드
배양액
발효기의 접종 전에 2-단계 씨드 트레인을 선택하였다. 제1 씨드 배양액은 새로 성장시킨 플레이트로부터 출발하여, 4개의 배플 (홈)을 갖는 500 mL 진탕 플라스크 내에서 100 mL의 배지를 접종함으로써 제조하였다. 배지는 20 g/L 글루코스를 갖는 SC-His-Leu-Ura-배지 (포미디엄, 영국 헌스탄톤)로 이루어졌다. 진탕 플라스크를 30℃에서 160 rpm에서 진탕 테이블 상에 놓았다. 세포를 약 0.5의 OD600까지 지수기로 성장시켰고, 이것은 글루코스 고갈 전이었다. 이어서, 제1 씨드 배양액을 사용하여, 0.025의 초기 OD600에 도달하도록 충분한 접종물을 이용하여 4개의 배플 (홈)을 갖는 500 mL 진탕 플라스크 내에서 제2 100 mL SC-His-Leu-Ura-배지 씨드 배양액을 제조하였다. 세포를 OD600이 3.2에 도달할 때까지 지수기로 성장시켰고, 이것은 글루코스 고갈 전이었다.
발효 배치기
발효는 1 L의 최대 작업 부피를 갖는 멀티포스 2 발효기 (인포스 아게, 스위스 모트밍겐) 내에서 수행하였다. 발효기에는 2개의 러시턴(Rushton) 6-블레이드 (blade) 및 4개 배플이 설치되었다. 공기를 이용하여 발효기에 살포하였다. 온도, pH, 교반, 및 통기 속도는 배양 내내 제어하였다. 온도는 20℃에서 유지하였다. pH는 5 M NH4OH 또는 0.5 M HCl의 자동 첨가에 의해 5.85로 유지하였다. 교반기 속도는 800 rpm으로 설정하고, 통기 속도는 1 vvm [L 기체/(L 액체 x min)]로 유지하였다. 발효는 0.32 L SC-His-Leu-Ura, 20 g/L 글루코스 및 0.01% 소포제 용액 (AF204, 시그마 (Sigma))의 출발 부피를 갖는, 38시간의 배치로서 출발하였다. 접종 전에, 발효기 내의 10 mL의 배지를 제거하였다. 이어서, 10 mL의 제2-단계 씨드 배양액 (상기 참조)의 분취액을 발효기의 접종을 위해 사용하여 0.32 L의 최종 부피를 얻었다. 따라서, 접종비는 3% (v/v)이었고, 이에 의해 0.1의 출발 OD600을 얻는다.
공급물
조성 및 일반적인 조건
38 hr의 배치 발효 시간 후에, 글루코스, 비타민, 미량-금속 및 염의 혼합물로 공급을 개시하고, 아미노산을 추가로 보충하였다 (표 52, 53 및 54 참조).
<표 52>
<표 53>
<표 54>
상기 공급기 동안, 수산화암모늄 (5 M NH4OH)을 질소원으로서 및 pH를 제어하기 위해 염기로서 사용하였다. pH를 제어하기 위해 사용한 산은 0.5 M HCl이었다. 공기를 사용하여 생물반응기에 살포하였다. 온도, pH, 교반, 및 통기 속도는 배양 내내 제어하였다. 온도는 20℃에서 유지하였다. pH는 5 M NH4OH 또는 0.5 M HCl의 자동 첨가에 의해 5.85로 유지하였다. 교반기 속도는 초기에 800 rpm으로 설정하고, 통기 속도를 초기에 1.0 vvm으로 설정하였다. 교반기 속도는 최대 1200 rpm에 도달하는 시간 스케줄 (아래 참조)에 따라, 수동으로 단계별로 증가시켰다. 통기는 부피 및 바이오매스 증가 동안 충분한 통기 속도를 유지하기 위해 수동으로 단계별로 증가시켰다 (아래 참조). 통기 및 교반의 관리는 용존 산소 (DO)가 10-20% 미만으로 하락하는 것을 방지하는 것을 목표로 하였다. 유가식 시기 동안 일반적인 작동 조건은 다음과 같았다: 액체 부피: 0.32 - 0.755 L, 온도 20℃, pH 5.85, 교반 속도 800-1200 rpm, 및 공기 유속 1-2 vvm.
공급물의 예상된 총 첨가는 아래에 개략한 공급 방식에 따라 72 hr 내에 435.6 mL이었다. 기체의 정체는 과도하지 않았지만, 여전히 실제 부피를 10%만큼 많이 증대시키는 것으로 예상되었다. 거품 및 기체-정체의 형성은 유가식 배양의 초기 배치기에 사용된 "SC-His-Leu-Ura" 배지 내로 소포제 첨가에 의해 제어하였다. 샘플을 1일 2회 채취하고, CCL 생산에 대해 분석하였다. 바이오매스의 증가에는 주로 OD600의 증가가 따라 일어났다.
공급 방식
공급기를 위해, 미리 정해진 지수적 공급 방식을 사용하였고, 이것은 임의의 유형의 설정값, 예컨대 용존 산소 등에 의해 제어되지 않았다. 지수적 공급 프로파일을 표 55에 제시하고, 교반 및 통기 속도를 표 56에 제시한다.
<표 55>
<표 56>
분석 절차 :
샘플 제조: 발효 동안 취한 효모 샘플을 10분 동안 1000 x g에서 원심분리하였다. 펠렛 및 상청액을 분리하였다. 추가로 정제하지 않고, 5 ㎕의 상청액을 micrOTOF-Q II (브루커 달토닉 게엠베하, 독일 브레멘)에 결합된 UPLC-TOF (워터스 액퀴티™ 초고성능 LC, 워터스, 미국 매사추세츠주 밀포드) 내에 주입하였다. 정지상: 컬럼은 액퀴티 UPLC® 브릿지드 에틸 하이브리드 (BEH) C18 1.7 ㎛ 2.1x100 mm이었다. 액체 크로마토그래피 방법: 이동상 A: H2O + 0.1% 포름산. 이동상 B: 아세토니트릴 + 0.1% 포름산. 진행 조건:
<표 57>
PDA 파라미터: λ 범위: 210 nm 내지 500 nm. 분리능: 1.2 nm. 샘플링 속도: 5 지점/sec. ELSD 파라미터: 게인: 50, 기체압: 40 psi, 네뷸라이저 방식: 가열, 파워 수준: 80%, 드리프트 튜브: 80℃. TOF 파라미터: 전원: 엔드 플레이트 오프셋: -500 V. 모세관: -4500 V. 네뷸라이저: 1.6 bar. 건조 기체: 8.0 L/min. 건조 온도: 180℃. 스캔 방식: MS 스캔. 질량 범위: 80 내지 1000 m/z.
결과:
CCL 및 페스투클라빈 (FCL)의 생산을 측정하였고, 화합물 역가/시간을 세포 밀도 (OD600)에 대해 그래프로 작성하였다 (도 26 참조). 이것은 세포 질량의 증가와 상호관련된 두 화합물 모두에서 증가를 보여주었다. 바이오매스의 증가는 울트로스펙 (Ultrospec) 10 세포 밀도계 (아머샴 바이오사이언시스 (Amersham Biosciences))에서 OD600을 0.49의 팩터를 곱하여 건조 세포량 (CDW)으로 전환시킴으로써 추정하였다.
청정화된 브로쓰 내에서 측정된 (즉, 세포의 제거 후에), 상기 공정으로 얻어진 CCL의 농도는 약 789 mg/L이었다 (도 26 참조). CCL의 분획이 세포 내에서 또한 검출되었지만, 여전히 대부분의 CCL을 청정화된 브로쓰로부터 단리할 수 있었다. 또한, 약 114 mg/L의 FCL이 생산되었다.
요약하면, 결과는 제어된 유가식 발효 공정 동안 EYS2325의 세포 질량을 증가시키면 CCL 및 FCL의 생산을 증가시킴을 보여준다.
실시예 15: 상이한 유전적 배경을 갖는 효모 균주 내에서 시클로클라빈의 생산.
물질 및 방법 :
본 실험에서 사용한 효모 균주를 표 58에 나열한다:
<표 58>
실시예 15에서 사용한 유전자를 표 59에 나열한다:
<표 59>
유전자의 클로닝 :
효모 내에서 발현을 위해 코돈 최적화된 합성 유전자. 유전자는 아미노산 서열 (서열 123, 및 102 내지 109) + 번역 중지 코돈을 코딩한다. 합성 동안, 모든 유전자에는 5'-단부에 HindIII 제한 인식 부위를 포함하는 DNA 서열 AAGCTTAAA, 및 3'-단부에 SacII 제한 부위를 포함하는 DNA 서열 CCGCGG이 제공되었다. 유전자를 HindIII 및 SacII를 사용하여, HindIII 및 SacII를 포함하는 다중의 클로닝 부위에 의해 분리된 천연 효모 프로모터 및 종결인자를 함유하는 효모 발현 카세트 (서열 110, 111, 124, 및 172) 내로 클로닝하였다. 모든 카세트에는 AscI 제한 부위가 측면에 접해있었다. 유전자 발현 카세트 (서열 110, 111, 124, 및 172)는 발현 카세트의 클로닝을 위한 AscI 부위를 제공하기 위해 링커 (서열 113)가 그 내부에 삽입된 pUC18 기반 플라스미드 벡터 (서열 112) 상에 포함되었다.
유전자 발현 카세트를 pUC18 기반 재조합 벡터 내로 서브-클로닝하였고, 이들은 각각 중심에서 AscI 부위 및 Mlu I 부위가 측면에 접해 있는, 120 bp 상동성 재조합 태그 (HRT) 서열을 가졌다. 이들 HRT는 제1 태그의 제2 절반이 제2 태그의 제1 절반에 동일하고 그 등등이 되도록 설계되었다.
3개의 헬퍼 단편을 사용하여 2개의 플라스미드를 조립하였고, 이들은 함께 전체 CCL 경로를 구성하였다: 1개의 헬퍼 단편은 효모 영양요구성 마커 및 박테리아 pMB1 복제 기원을 포함하였다. 제2 헬퍼 단편은 효모 내에서 복제를 위한 ARS4/CEN6 서열 및 박테리아 클로람페니콜 내성 마커를 포함하였다. 두 단편은 측면 HRT를 가졌다. 제3 단편은 짧은 600 bp 스페이서 서열에 의해 분리된 HRT만을 사용하여 설계되었다. 모든 헬퍼 단편은 이. 콜라이 내에서 증폭을 위해 pUC18 기반 백본 내에 클로닝되었다. 모든 단편은 그들이 절제될 수 있는 AscI 부위 내에 클로닝되었다.
2개의 플라스미드 CCL-HRT1 및 CCL-HRT5 (서열 173 및 174)를 제조하기 위해, 3개의 헬퍼 플라스미드로부터 플라스미드 DNA를 CCL-HRT1에 대해 CpDmaW, AfEasF, AjEasE, 및 AjEasC, 또는 CCL-HRT5에 대해 AjEasD, AjEasG, AjEasH, 및 AjEasA를 포함하는 플라스미드 DNA와 혼합하였다. 플라스미드 DNA의 혼합물을 AscI로 소화시켰다. 이것은 모든 단편을 플라스미드 백본으로부터 방출시키고, 말단에서 HRT를 갖는 단편을 생산하고, 이것은 다음 단편의 HRT와 순차적으로 겹친다. 효모 균주 EYS1456을 각각의 소화된 혼합물로 형질전환시키고, 플라스미드 CCL-HRT1 및 CCL-HRT5를 문헌 [Shao 2009]에 설명된 바와 같이 상동성 재조합에 의해 생체 내에서 조립하였다. 2개의 플라스미드를 효모 형질전환체로부터 단리하고, 플라스미드 DNA의 용이한 증폭 및 정제를 허용하는 이. 콜라이 내로 역 형질전환시켰다. 2개의 플라스미드 CCL-HRT1 및 CCL-HRT5를 효모 균주의 패널 내로 동시-형질전화시켰다 (표 58).
성장 조건 :
2개의 벡터 CCL-HRT1 및 CCL-HRT5를 포함하는 조작된 효모 균주를 류신 및 우라실이 없는, 2% 글루코스를 갖는 3 mL 표준 SC 브로쓰 (포미디엄, 영국 헌스탄톤) 내에서 성장시켰다. 배양액을 20℃에서 96시간 동안 연속적으로 진탕하면서 성장시켰다.
분석 절차 :
샘플 제조: 효모 배양액을 10분 동안 1000 x g에서 원심분리하였다. 펠렛 및 상청액을 분리하였다. 추가로 정제하지 않고, 5 ㎕의 상청액을 micrOTOF-Q II (브루커 달토닉 게엠베하, 독일 브레멘)에 결합된 UPLC-TOF (워터스 액퀴티™ 초고성능 LC, 워터스, 미국 매사추세츠주 밀포드) 내에 주입하였다. 정지상: 컬럼은 액퀴티 UPLC® 브릿지드 에틸 하이브리드 (BEH) C18 1.7 ㎛ 2.1x100 mm이었다. 액체 크로마토그래피 방법: 이동상 A: H2O + 0.1% 포름산. 이동상 B: 아세토니트릴 + 0.1% 포름산. 진행 조건:
<표 60>
PDA 파라미터: λ 범위: 210 nm 내지 500 nm. 분리능: 1.2 nm. 샘플링 속도: 5 지점/sec. ELSD 파라미터: 게인: 50, 기체압: 40 psi, 네뷸라이저 방식: 가열, 파워 수준: 80%, 드리프트 튜브: 80℃. TOF 파라미터: 전원: 엔드 플레이트 오프셋: -500 V. 모세관: -4500 V. 네뷸라이저: 1.6 bar. 건조 기체: 8.0 L/min. 건조 온도: 180℃. 스캔 방식: MS 스캔. 질량 범위: 80 내지 1000 m/z.
결과 :
모든 균주의 배양 상청액을 LC-MS에 의해 분석하고, 예상된 질량의 시클로클라빈 (m/z = 239.1543+/-0.01 Da) 및 예상된 질량의 페스투클라빈 (m/z = 241.1699+/-0.01 Da)의 이온 크로마토그램을 추출하였다. 피크 하 면적을 통합하고, CCL 및 FCL의 생산을 내부 표준물에 기반하여 계산하였다. CCL 및 FCL의 양을 비교하였고 (도 27), 이것은 모든 균주 내에서 두 화합물의 생산을, 그러나 다양한 양으로의 생산을 보여주었다. 이들 결과는 CCL 및 FCL 생산이 에스. 세레비지아에의 특이적 균주 또는 유전자형에 의존하지 않음을 보여준다.
참고문헌:
SEQUENCE LISTING
<110> BASF SE
Evolva SA
<120> Genes and Processes for the Production of Clavine-Type Alkaloids
<130> PF 74187
<150> US 61/691848
<151> 08-22-2012
<150> EP 12181388.5
<151> 08-22-2012
<150> US 61/786841
<151> 03-15-2013
<150> EP 13159444.2
<151> 03-15-2013
<150> EP 13178008.2
<151> 07-25-2013
<160> 192
<170> PatentIn version 3.5
<210> 1
<211> 534
<212> PRT
<213> Aspergillus japonicus
<400> 1
Met Thr Ala Gly Gln Gly Ile Lys Thr Gly Asn Ala Ser Asp Cys Glu
1 5 10 15
Val Tyr Arg Thr Leu Ser Val Ala Leu Asp Phe Ala Asn Gln Asp Glu
20 25 30
Glu Leu Trp Trp His Ser Thr Ala Pro Met Phe Ala Gln Met Leu Gln
35 40 45
Ser Thr Asn Tyr Asn Leu His Ala Gln Tyr Lys His Leu Leu Ile Tyr
50 55 60
Lys Lys Asn Val Ile Pro Phe Leu Gly Val Tyr Pro Thr Asn Asp Lys
65 70 75 80
Pro Arg Trp Leu Ser Ile Leu Thr Arg Tyr Gly Thr Pro Phe Glu Leu
85 90 95
Ser Leu Asn Cys Ser Gly Pro Leu Val Arg Tyr Thr Tyr Glu Pro Ile
100 105 110
Asn Ala Ala Thr Gly Thr Ala Arg Asp Pro Phe Asn Thr His Ala Val
115 120 125
Trp Asp Ser Leu Glu Gln Leu Met Ala Leu Gln Ser Gly Ile Asp Leu
130 135 140
Asp Leu Phe Arg His Phe Lys Asn Asp Leu Thr Leu Ser Ala Glu Glu
145 150 155 160
Ser Glu Tyr Leu Tyr Lys Asn Asn Leu Val Gly Glu Gln Ile Arg Thr
165 170 175
Gln Asn Lys Leu Ala Leu Asp Leu Gln Asp Gly Glu Phe Val Val Lys
180 185 190
Thr Tyr Ile Tyr Pro Ala Leu Lys Ser Leu Ala Thr Gly Arg Ser Ile
195 200 205
His Glu Leu Val Phe Gly Ser Ala Phe Arg Trp Ser Lys Gln Tyr Pro
210 215 220
Glu Leu Arg Lys Pro Leu Asp Thr Leu Glu Gln Tyr Val Tyr Ser Arg
225 230 235 240
Gly Pro Ser Ser Thr Ala Ser Pro Arg Leu Leu Ser Cys Asp Leu Ile
245 250 255
Asp Pro Thr Lys Ser Arg Ile Lys Ile Tyr Leu Leu Glu Arg Met Val
260 265 270
Thr Leu Glu Ala Leu Glu Asp Leu Trp Thr Met Gly Gly Glu Arg Thr
275 280 285
Asp Ala Ser Thr Leu Ala Gly Leu Glu Met Ile Arg Glu Leu Trp Glu
290 295 300
Leu Ile Arg Leu Pro Ala Gly Leu Gln Ser Tyr Pro Ala Pro Tyr Leu
305 310 315 320
Pro Ile Gly Thr Ile Pro Asp Glu Gln Leu Pro Leu Met Ala Asn Tyr
325 330 335
Thr Ile His His Asp Asp Pro Val Pro Glu Pro Gln Val Tyr Phe Thr
340 345 350
Thr Phe Gly Arg Asn Asp Met Gln Ile Ala Asp Ala Leu Ala Thr Phe
355 360 365
Phe Glu Arg Arg Gly Trp His Glu Met Ala Arg Gln Tyr Lys Ala Glu
370 375 380
Leu Cys Ser His Tyr Pro His Ala Asp His Glu Thr Leu Asn Tyr Leu
385 390 395 400
His Ala Tyr Ile Ser Phe Ser Tyr Arg Lys Asn Lys Pro Tyr Leu Ser
405 410 415
Val Tyr Leu Gln Ser Leu Glu Thr Gly Asp Trp Val Thr Cys Lys Leu
420 425 430
Ser Pro Ser Val Phe Leu Ser Cys Thr Leu Pro His Arg Gly Tyr Ile
435 440 445
Pro Asp Asp Leu Thr Cys Ser Arg Ser Ile Leu Gln Leu Cys Pro Cys
450 455 460
Gly Pro Arg Pro Leu Ser His Arg Pro Arg Ala Val Gln Ala Asp Gln
465 470 475 480
Asp Arg Arg Asp His Arg Ser Arg Asp Glu Thr Pro Thr His Pro Gly
485 490 495
Trp Ile Gly Ala Arg Arg His His Pro Val Leu Gly Asn His Leu Val
500 505 510
Gly Pro Asp Leu Pro Ala Ser Arg Arg Gly Val Ala Ser Ala Leu Tyr
515 520 525
Val Gly Arg Gly Thr Arg
530
<210> 2
<211> 412
<212> PRT
<213> Aspergillus japonicus
<400> 2
Met Thr Ile Asp Pro Ser Ser Pro Gly Ala Pro Ala Ser Lys Leu Phe
1 5 10 15
Gln Pro Met Lys Ile Gly Leu Cys His Leu Gln His Arg Leu Ile Met
20 25 30
Ala Pro Thr Thr Arg Tyr Arg Ala Asp Ser Thr Ala Thr Pro Leu Pro
35 40 45
Phe Val Gln Glu Tyr Tyr Ala Gln Arg Ala Ala Val Pro Gly Thr Leu
50 55 60
Leu Ile Thr Glu Ala Thr Asp Ile Ser Pro Gln Ala Ala Gly Glu Pro
65 70 75 80
His Ile Pro Gly Leu Tyr Ser Ala Thr Gln Ile Ala Ala Trp Arg Gly
85 90 95
Ile Val Ser Ala Val His Arg Arg Gly Cys Tyr Ile Phe Cys Gln Leu
100 105 110
Trp Ala Thr Gly Arg Ala Ala Asp Pro Ala Leu Leu Lys Glu Lys Gly
115 120 125
Phe Pro Leu Val Ser Ser Ser Asp Val Pro Ile Glu Thr Glu Ser Gln
130 135 140
Gln Pro Gln Pro Leu Thr Gln Ala Glu Ile Glu Gly Tyr Ile Ala Asp
145 150 155 160
Phe Val Thr Ala Ala Arg Val Ala Val His Glu Val Gly Phe Asp Gly
165 170 175
Val Glu Leu His Gly Ala Asn Gly Tyr Leu Ile Asp Gln Phe Thr Gln
180 185 190
Trp Pro Cys Asn Gln Arg Thr Asp Cys Trp Gly Gly Ser Ile Glu Asn
195 200 205
Arg Ala Arg Phe Ala Leu Ala Val Thr Arg Ala Val Val Asp Ala Val
210 215 220
Gly Ala Glu Arg Val Gly Val Lys Leu Ser Pro Trp Ser Gln Tyr Ala
225 230 235 240
Gly Met Gly Val Met Ala Asp Leu Val Pro Gln Phe Glu Phe Leu Ile
245 250 255
Lys Arg Leu Arg Glu Glu Tyr Asn Asp Gly Pro Gly Leu Ala Tyr Leu
260 265 270
His Leu Ala Asn Ser Arg Trp Leu Asp Glu Glu Thr Ser His Pro Asp
275 280 285
Pro His His Glu Val Phe Val Arg Ala Trp Arg Gly Ser Gly Arg Asp
290 295 300
Gly Ile Glu Ala Lys Lys Glu Ala Pro Pro Val Ile Leu Ala Gly Gly
305 310 315 320
Tyr Asp Ala Val Ser Ala Pro Glu Val Ala Asp Lys Val Phe Lys Glu
325 330 335
Glu Asp Asn Val Ala Val Ala Phe Gly Arg Tyr Phe Ile Ser Thr Pro
340 345 350
Asp Leu Pro Phe Arg Met Gln Arg Gly Ile Pro Leu Gln Lys Tyr Asp
355 360 365
Arg Ala Ser Phe Tyr Thr Ser Leu Leu Lys Glu Gly Tyr Leu Asp Tyr
370 375 380
Pro Tyr Ser Ala Glu Tyr Val Ala Glu Phe Gly Gln Arg Leu Lys Lys
385 390 395 400
Lys Glu Ala Ala Ala Thr Gly Ser Ile Cys Gln Gly
405 410
<210> 3
<211> 509
<212> PRT
<213> Aspergillus japonicus
<400> 3
Met Ala Pro Asp Ser Lys Pro Thr Tyr Thr Thr Ala Asn Gly Cys Pro
1 5 10 15
Phe His Lys Pro Glu Gly Pro Pro Ala Asp Gly Arg Thr Leu Ala Leu
20 25 30
Ser Asp His His Leu Val Asp Thr Leu Ala His Phe Asn Arg Glu Lys
35 40 45
Ile Pro Glu Arg Val Val His Ala Lys Gly Ala Gly Ala Tyr Gly Glu
50 55 60
Phe Glu Val Thr Ala Asp Ile Ser Asp Ile Cys Asp Ile Asp Met Leu
65 70 75 80
Val Gly Val Gly Lys Lys Thr Pro Cys Val Thr Arg Phe Ser Thr Thr
85 90 95
Gly Leu Glu Arg Gly Ser Asn Glu Gly Met Arg Asp Leu Lys Gly Met
100 105 110
Ala Cys Lys Phe Tyr Thr Thr Glu Gly Asn Trp Asp Trp Val Met Leu
115 120 125
Asn Phe Pro Phe Phe Phe Ile Arg Asp Pro Val Lys Phe Pro Ser Leu
130 135 140
Met His Ala Gln Arg Arg Asp Pro Gln Thr Asn Leu Leu Asn Pro Asn
145 150 155 160
Met Tyr Trp Asp Trp Val Thr Asn Asn His Glu Ser Leu His Met Val
165 170 175
Leu Leu Gln Phe Ser Asp Phe Gly Thr Met Phe Asn Trp Arg Ser Met
180 185 190
Ser Gly Tyr Met Ala His Ala Tyr Lys Trp Val Lys Pro Asp Gly Ser
195 200 205
Phe Lys Tyr Val His Ile Phe Leu Ser Ser Asp Arg Gly Pro Asn Phe
210 215 220
Thr Asp Gly Gln Gln Ala Lys Gly Thr Asn Asp Leu Asp Pro Asp His
225 230 235 240
Ala Thr Arg Asp Leu Tyr Glu Ala Ile Glu Arg Gly Glu Tyr Pro Thr
245 250 255
Trp Thr Ala Ser Val Gln Val Val Asp Pro Lys Asp Ala Pro Asn Leu
260 265 270
Gly Tyr Asn Ile Leu Asp Val Thr Lys His Trp Asn Leu Gly Thr Tyr
275 280 285
Pro Lys Asp Val Thr Leu Ile Pro Pro Lys Val Phe Gly Lys Leu Thr
290 295 300
Leu Lys Arg Asn Pro Ala Asn Tyr Phe Ala Glu Ile Glu Gln Leu Ala
305 310 315 320
Tyr Ser Pro Ser Asn Met Val Pro Gly Val Ala Pro Ser Glu Asp Pro
325 330 335
Ile Leu Gln Ala Arg Ile Phe Ala Tyr Pro Asp Ala Gln Arg Tyr Arg
340 345 350
Leu Gly Ala Asn His Gln Gln Ile Pro Val Asn Arg Ser Ala His Thr
355 360 365
Phe Asn Pro Ile Ala Arg Asp Gly Gln Gly Thr Phe Asp Ala Asn Tyr
370 375 380
Gly Ala His Pro Gly Phe Leu Thr Gln Gln Gln Pro Val Arg Phe Ala
385 390 395 400
Glu Pro Arg Glu Pro Asp Pro Lys Tyr Asn Glu Trp Leu Lys Glu Ile
405 410 415
Gln Ser Lys Ser Trp Leu Gln Thr Thr Glu His Asp Tyr Lys Phe Ala
420 425 430
Arg Asp Phe Tyr Glu Val Leu Pro Asp Phe Arg Gly Gln Glu Phe Gln
435 440 445
Asp Thr Met Val Gln Asn Met Val Glu Ser Val Ala Gln Thr Arg Ala
450 455 460
Glu Ile Gln Lys Gln Val Tyr Glu Thr Trp Lys Leu Val Ser Pro Ala
465 470 475 480
Leu Ala Ala Arg Ile Gln Lys Gly Val Glu Thr Leu Leu Gln Lys Ser
485 490 495
Glu Ala Pro Ala Glu Glu Ser Phe Ile Pro Ala Arg Leu
500 505
<210> 4
<211> 262
<212> PRT
<213> Aspergillus japonicus
<400> 4
Met Thr Val Val Asn Ser Arg Ile Phe Ala Val Thr Gly Gly Ala Ser
1 5 10 15
Gly Ile Gly Ala Ala Thr Cys Arg Leu Leu Ala Ala Arg Gly Ala Ala
20 25 30
Val Ile Cys Ile Gly Asp Ile Ser Pro Lys Gly Phe Glu Ser Val Thr
35 40 45
Ser Asp Ile Gln Ala Leu Asn Pro Ala Thr Lys Val His Cys Thr Arg
50 55 60
Leu Asp Val Ser Ser Ser Ala Glu Val Asp Ala Trp Leu Gln Thr Ile
65 70 75 80
Val Gln Thr Phe Gly Asp Leu His Gly Leu Ala Asn Val Ala Gly Ile
85 90 95
Ala Gln Pro Ala Gly Ala Arg Gln Ala Pro Ala Leu Leu Glu Glu Thr
100 105 110
Asp Glu Glu Trp Gln Arg Val Phe Lys Val Asn Leu Asp Gly Val Phe
115 120 125
Tyr Cys Thr Arg Ala Gly Ile Arg Ala Met Lys Ala Leu Pro Gly Pro
130 135 140
Lys Asp Arg Ala Ile Val Asn Ile Gly Ser Ile Ala Ala Phe Ile His
145 150 155 160
Leu Pro Asp Ala Tyr Ala Tyr Gly Thr Ser Lys Gly Ala Cys Ala Tyr
165 170 175
Phe Thr Ser Cys Ala Ala Met Asp Ala Trp Pro Leu Gly Ile Arg Ile
180 185 190
Asn Ser Val Ser Pro Gly Ile Thr Asp Thr Pro Ala Leu Pro Gln Phe
195 200 205
Met Pro Gln Ala Lys Ser Ile Asp Glu Val Lys Glu Ala Tyr Ile Lys
210 215 220
Glu Gly Phe Pro Ile Ile Gln Pro Glu Asp Val Ala Arg Thr Ile Val
225 230 235 240
Trp Leu Leu Ser Glu Asp Ser Arg Pro Val Tyr Gly Ala Asn Ile Asn
245 250 255
Val Gly Ala Ala Thr Pro
260
<210> 5
<211> 621
<212> PRT
<213> Aspergillus japonicus
<400> 5
Met Gly Gln Ser Arg Gly Ile Leu Gly Gly Val Arg Gln Leu Ile Leu
1 5 10 15
Val Ile Leu Val Gly Ala Tyr Leu Ser Arg Leu Ser Ala Val Asp Asp
20 25 30
Asp Arg His Asp Cys Arg Cys Arg Pro Gly Glu Pro Cys Trp Pro Thr
35 40 45
Val Ser His Trp Ser Thr Leu Asn Glu Ser Ile Gly Gly Ala Leu Ala
50 55 60
His Val Lys Pro Ile Ala His Val Cys His Gln Ser Gly Gln Asp Ser
65 70 75 80
Ser Ala Cys Glu Gln Val Leu Gln Glu Ser Leu Asp Ser Lys Trp Arg
85 90 95
Ala Ser His Thr Gly Ala Leu Gln Asp Trp Val Trp Glu Gly Gly Ala
100 105 110
Glu Ser Asn Gln Thr Cys Tyr Tyr Leu Arg Ser Gly Pro Ala Gly Ser
115 120 125
Cys His Gln Gly Arg Ile Pro Leu Tyr Ser Ala Ala Val Lys Ser Ala
130 135 140
Ser Asp Val Gln Lys Val Val Asp Phe Thr Arg Gln His Asn Leu Arg
145 150 155 160
Leu Val Ile Arg Asn Thr Gly His Asp Gly Ser Gly Arg Ser Ser Gly
165 170 175
Pro Asp Ser Val Glu Ile His Thr His His Leu Asn Ser Val Gln Tyr
180 185 190
His Pro Asn Phe Arg Pro Ala Gly Ser Ser Glu Arg Gln Ser Ala Pro
195 200 205
Gly Gln Pro Ala Val Thr Val Gly Ala Gly Ile Leu Leu Gly Asp Leu
210 215 220
Tyr Ala Arg Gly Ala Ser Glu Gly Trp Ile Val Val Gly Gly Glu Cys
225 230 235 240
Pro Thr Val Gly Ala Ala Gly Gly Phe Leu Gln Gly Gly Gly Val Ser
245 250 255
Ser Trp Leu Ser Tyr Ala His Gly Leu Ala Val Asp Asn Val Leu Glu
260 265 270
Tyr Glu Val Val Thr Ala Lys Gly Glu Ile Val Ile Ala Asn Ala His
275 280 285
Gln Asn Pro Asp Leu Phe Trp Ala Leu Arg Gly Gly Gly Gly Gly Thr
290 295 300
Phe Gly Val Val Thr Gln Ala Thr Leu Gln Val His Pro Asp Leu Pro
305 310 315 320
Val Ser Val Ala Asp Ala Val Val Thr Gly Ser Arg Ala Asp Ala Thr
325 330 335
Phe Trp Ser His Gly Val Ala Ala Leu Leu Arg Ala Leu Gln Phe Leu
340 345 350
Asn Asn His Gly Thr Ala Gly Gln Phe Ile Leu Arg Asn Thr Asp Asp
355 360 365
Asp Thr Val Gln Ala Ser Leu Thr Met Tyr Phe Ser Asn Leu Thr Val
370 375 380
Pro Ala Val Ala Asp Glu Arg Met Asp Pro Leu Arg Arg Ala Leu Glu
385 390 395 400
His Asn Gly His Pro Tyr Gln Leu Thr Ser Arg Phe Leu Pro Gln Ile
405 410 415
Ser Ser Thr Phe Arg His Thr Ala Asp Arg Tyr Pro Glu Asp Tyr Gly
420 425 430
Ile Leu Met Gly Ser Val Leu Val Ser Leu Asp Leu Phe Asn Ser Ala
435 440 445
Thr Gly Pro Ala Ala Leu Ala Gln His Phe Ala Arg Leu Pro Met Thr
450 455 460
Pro Asp Asp Leu Leu Phe Thr Ser Asn Leu Gly Gly Arg Val Ser Ser
465 470 475 480
Leu Asn Arg Asp Pro Ala Ser Thr Ala Met His Pro Gly Trp Arg Asp
485 490 495
Ala Ala Gln Leu Leu Asn Phe Val Arg Gly Val Gly Ala Pro Ser Leu
500 505 510
Ala Ala Lys Ala Thr Ala Leu His Glu Leu Gln Thr Val Gln Met Ala
515 520 525
Arg Leu Tyr Glu Ile Glu Pro Ala Phe Gln Ile Ser Tyr Arg Asn Leu
530 535 540
Gly Asp Pro Ser Glu Arg Arg Ser Arg Glu Val Tyr Trp Gly Ser Asn
545 550 555 560
Tyr Ala Arg Leu Val Glu Val Lys Arg Arg Trp Asp Pro Glu Gly Leu
565 570 575
Phe Phe Ser Lys Leu Gly Ile Asp Gly Asp Ala Trp Asp Ala Glu Gly
580 585 590
Met Cys Arg Gln Thr Arg Gln Gly Ala Trp Asn Val Ala Val Lys Trp
595 600 605
Val Gln Ser Phe Leu Gly Ser Leu Ser Ala Ser Val Val
610 615 620
<210> 6
<211> 339
<212> PRT
<213> Aspergillus japonicus
<400> 6
Met Gly Ser Ile Asn Pro Pro Ile Leu Asp Ile Arg Gln Ser Thr Phe
1 5 10 15
Glu Thr Ser Ile Pro Gln Gln Val Thr Glu Gly Leu Thr Lys Asp Pro
20 25 30
Lys Thr Leu Pro Ala Leu Leu Phe Tyr Ser Gly Asp Gly Ile Gln His
35 40 45
Trp Thr Arg His Ser Cys Ala Pro Asp Phe Tyr Pro Arg Arg Glu Glu
50 55 60
Ile Asn Ile Leu His Ser Glu Ala Ala His Met Ala Ala Ser Ile Ser
65 70 75 80
Gln Asp Ser Val Val Val Asp Leu Gly Ser Ala Ser Leu Asp Lys Val
85 90 95
Leu Pro Phe Leu His Ala Leu Glu Ala Gln Gln Lys Arg Val His Tyr
100 105 110
Tyr Ala Leu Asp Leu Ser Tyr Pro Val Leu Ala Ser Thr Leu Ser Glu
115 120 125
Ile Pro Val Ala Gln Phe Gln Tyr Val Gln Ile Ala Ala Leu His Gly
130 135 140
Thr Phe Glu Asp Gly Leu Gln Trp Leu His Asp Ser Pro Glu Val Arg
145 150 155 160
Glu Arg Pro His His Leu Leu Leu Phe Gly Leu Thr Ile Gly Asn Phe
165 170 175
Ser Arg Pro Asn Ala Ala Ala Phe Leu Arg Asn Ile Ala Asp Arg Ala
180 185 190
Leu Ala Ala Ser Pro Ala Glu Ser Ser Ile Leu Leu Thr Leu Asp Ser
195 200 205
Cys Lys Met Pro Thr Lys Val Leu Arg Ala Tyr Thr Ala Glu Gly Val
210 215 220
Val Pro Phe Ala Leu Thr Ser Leu Ser Tyr Ala Asn Gln Leu Phe Trp
225 230 235 240
Pro Gly Gly Glu Gly Arg Val Phe Asp Glu Glu Glu Trp Tyr Phe His
245 250 255
Ser Glu Trp Asn Phe Ala Leu Gly Arg His Glu Ala Ala Leu Ile Pro
260 265 270
Arg Asp Arg Asp Ile Thr Leu Gly Pro Pro Leu Glu Gln Val Val Val
275 280 285
Arg Arg Gly Glu Lys Val Arg Phe Gly Cys Ser Tyr Lys Tyr Ser Ala
290 295 300
Ala Glu Arg Glu His Leu Phe Ala Gly Ala Gly Leu Glu Asn Val Ala
305 310 315 320
Ser Trp Ser Ala Lys Gly Cys Asp Val Ala Phe Tyr Gln Leu Lys Met
325 330 335
Arg Pro Glu
<210> 7
<211> 284
<212> PRT
<213> Aspergillus japonicus
<400> 7
Met Thr Ile Leu Val Leu Gly Gly Arg Gly Lys Thr Ala Ser Arg Leu
1 5 10 15
Ser Ala Leu Leu His Glu Ala Gln Ile Pro Phe Val Val Gly Thr Ser
20 25 30
Ser Thr Ala Pro Ile Ala Thr Tyr Arg Thr Ala His Phe Asp Trp Leu
35 40 45
Asn Lys Asp Thr Trp Thr Asn Ala Trp Asp Gln Ala Pro Glu Pro Ile
50 55 60
Thr Ala Ile Tyr Leu Val Gly Ala His Ala Pro Glu Phe Val His Pro
65 70 75 80
Met Phe Glu Phe Ile Asp Leu Ala His Ser Arg Gly Val Arg Arg Tyr
85 90 95
Ala Leu Val Ser Ala Ser Asn Thr Glu Lys Gly Asp Pro Met Met Gly
100 105 110
Glu Ala His Ala His Leu Asp Ser Lys Glu Gly Val Lys Tyr Val Val
115 120 125
Leu Arg Pro Thr Trp Phe Met Glu Asn His Ile Glu Asp Pro His Leu
130 135 140
Ser Trp Ile Lys Thr Glu Asn Lys Ile Tyr Ser Ala Thr Gly Pro Gly
145 150 155 160
Lys Ile Pro Phe Val Ser Ala Asp Asp Ile Ala Arg Val Ala Phe His
165 170 175
Ala Leu Thr Gln Pro Leu Thr Gln Lys Glu Gln Gln Tyr Met Val Leu
180 185 190
Gly Pro Asp Leu Leu Ser Tyr Ala Asp Met Ala Glu Ile Leu Thr Asp
195 200 205
Leu Leu Gly Arg Lys Ile Thr His Val Thr Leu Pro Ala Glu Lys Phe
210 215 220
Gly Gln Phe Leu Val Asp Thr Ala Gly Leu Pro Pro Asp Phe Ala Glu
225 230 235 240
Met Leu Ala Thr Met Glu Ala Ala Val Ala Thr Gly Ala Glu Glu Arg
245 250 255
Val Ser Gln Asp Val Glu Lys Val Thr Gly Arg Pro Pro Arg Arg Phe
260 265 270
Val Asp Tyr Ala Arg Glu Asn Arg Glu His Trp Leu
275 280
<210> 8
<211> 293
<212> PRT
<213> Aspergillus japonicus
<400> 8
Met Thr Ile Thr Glu Thr Ser Lys Pro Thr Leu Arg Arg Ile Pro Arg
1 5 10 15
Ser Ala Gly Asp Glu Ala Ile Phe Gln Val Leu Gln Glu Asp Gly Val
20 25 30
Val Val Ile Glu Gly Phe Met Ser Ala Asp Gln Val Arg Arg Phe Asn
35 40 45
Gly Glu Ile Asp Pro His Met Lys Gln Trp Glu Leu Gly Gln Lys Ser
50 55 60
Tyr Gln Glu Ser Tyr Leu Ala Gly Met Arg Gln Leu Ser Ser Leu Pro
65 70 75 80
Leu Phe Ser Lys Leu Phe Arg Asp Glu Leu Met Asn Asp Glu Leu Leu
85 90 95
His Gly Leu Cys Lys Arg Leu Phe Gly Pro Glu Ser Gly Asp Tyr Trp
100 105 110
Leu Thr Thr Ser Ser Val Leu Glu Thr Glu Pro Gly Tyr His Gly Gln
115 120 125
Glu Leu His Arg Glu His Asp Gly Ile Pro Ile Cys Thr Thr Leu Gly
130 135 140
Arg Gln Ser Pro Glu Ser Met Leu Asn Phe Leu Thr Ala Leu Thr Asp
145 150 155 160
Phe Thr Ala Glu Asn Gly Ala Thr Arg Val Leu Pro Gly Ser His Leu
165 170 175
Trp Glu Asp Phe Ser Ala Pro Pro Pro Lys Ala Asp Thr Ala Ile Pro
180 185 190
Ala Val Met Asn Pro Gly Asp Ala Val Leu Phe Thr Gly Lys Thr Leu
195 200 205
His Gly Ala Gly Lys Asn Asn Thr Thr Asp Phe Leu Arg Arg Gly Phe
210 215 220
Pro Leu Ile Met Gln Ser Cys Gln Phe Thr Pro Val Glu Ala Ser Val
225 230 235 240
Ala Leu Pro Arg Glu Leu Val Glu Thr Met Thr Pro Leu Ala Gln Lys
245 250 255
Met Val Gly Trp Arg Thr Val Ser Ala Lys Gly Val Asp Ile Trp Thr
260 265 270
Tyr Asp Leu Lys Asp Leu Ala Thr Gly Ile Asp Leu Lys Ser Asn Gln
275 280 285
Val Ala Lys Lys Ala
290
<210> 9
<211> 440
<212> PRT
<213> Saccharomyces cerevisiae
<400> 9
Leu Gln Leu Ala Leu His Pro Val Glu Met Lys Ala Ala Leu Lys Leu
1 5 10 15
Lys Phe Cys Arg Thr Pro Leu Phe Ser Ile Tyr Asp Gln Ser Thr Ser
20 25 30
Pro Tyr Leu Leu His Cys Phe Glu Leu Leu Asn Leu Thr Ser Arg Ser
35 40 45
Phe Ala Ala Val Ile Arg Glu Leu His Pro Glu Leu Arg Asn Cys Val
50 55 60
Thr Leu Phe Tyr Leu Ile Leu Arg Ala Leu Asp Thr Ile Glu Asp Asp
65 70 75 80
Met Ser Ile Glu His Asp Leu Lys Ile Asp Leu Leu Arg His Phe His
85 90 95
Glu Lys Leu Leu Leu Thr Lys Trp Ser Phe Asp Gly Asn Ala Pro Asp
100 105 110
Val Lys Asp Arg Ala Val Leu Thr Asp Phe Glu Ser Ile Leu Ile Glu
115 120 125
Phe His Lys Leu Lys Pro Glu Tyr Gln Glu Val Ile Lys Glu Ile Thr
130 135 140
Glu Lys Met Gly Asn Gly Met Ala Asp Tyr Ile Leu Asp Glu Asn Tyr
145 150 155 160
Asn Leu Asn Gly Leu Gln Thr Val His Asp Tyr Asp Val Tyr Cys His
165 170 175
Tyr Val Ala Gly Leu Val Gly Asp Gly Leu Thr Arg Leu Ile Val Ile
180 185 190
Ala Lys Phe Ala Asn Glu Ser Leu Tyr Ser Asn Glu Gln Leu Tyr Glu
195 200 205
Ser Met Gly Leu Phe Leu Gln Lys Thr Asn Ile Ile Arg Asp Tyr Asn
210 215 220
Glu Asp Leu Val Asp Gly Arg Ser Phe Trp Pro Lys Glu Ile Trp Ser
225 230 235 240
Gln Tyr Ala Pro Gln Leu Lys Asp Phe Met Lys Pro Glu Asn Glu Gln
245 250 255
Leu Gly Leu Asp Cys Ile Asn His Leu Val Leu Asn Ala Leu Ser His
260 265 270
Val Ile Asp Val Leu Thr Tyr Leu Ala Gly Ile His Glu Gln Ser Thr
275 280 285
Phe Gln Phe Cys Ala Ile Pro Gln Val Met Ala Ile Ala Thr Leu Ala
290 295 300
Leu Val Phe Asn Asn Arg Glu Val Leu His Gly Asn Val Lys Ile Arg
305 310 315 320
Lys Gly Thr Thr Cys Tyr Leu Ile Leu Lys Ser Arg Thr Leu Arg Gly
325 330 335
Cys Val Glu Ile Phe Asp Tyr Tyr Leu Arg Asp Ile Lys Ser Lys Leu
340 345 350
Ala Val Gln Asp Pro Asn Phe Leu Lys Leu Asn Ile Gln Ile Ser Lys
355 360 365
Ile Glu Gln Phe Met Glu Glu Met Tyr Gln Asp Lys Leu Pro Pro Asn
370 375 380
Val Lys Pro Asn Glu Thr Pro Ile Phe Leu Lys Val Lys Glu Arg Ser
385 390 395 400
Arg Tyr Asp Asp Glu Leu Val Pro Thr Gln Gln Glu Glu Glu Tyr Lys
405 410 415
Phe Asn Met Val Leu Ser Ile Ile Leu Ser Val Leu Leu Gly Phe Tyr
420 425 430
Tyr Ile Tyr Thr Leu His Arg Ala
435 440
<210> 10
<211> 352
<212> PRT
<213> Saccharomyces cerevisiae
<400> 10
Met Ala Ser Glu Lys Glu Ile Arg Arg Glu Arg Phe Leu Asn Val Phe
1 5 10 15
Pro Lys Leu Val Glu Glu Leu Asn Ala Ser Leu Leu Ala Tyr Gly Met
20 25 30
Pro Lys Glu Ala Cys Asp Trp Tyr Ala His Ser Leu Asn Tyr Asn Thr
35 40 45
Pro Gly Gly Lys Leu Asn Arg Gly Leu Ser Val Val Asp Thr Tyr Ala
50 55 60
Ile Leu Ser Asn Lys Thr Val Glu Gln Leu Gly Gln Glu Glu Tyr Glu
65 70 75 80
Lys Val Ala Ile Leu Gly Trp Cys Ile Glu Leu Leu Gln Ala Tyr Phe
85 90 95
Leu Val Ala Asp Asp Met Met Asp Lys Ser Ile Thr Arg Arg Gly Gln
100 105 110
Pro Cys Trp Tyr Lys Val Pro Glu Val Gly Glu Ile Ala Ile Asn Asp
115 120 125
Ala Phe Met Leu Glu Ala Ala Ile Tyr Lys Leu Leu Lys Ser His Phe
130 135 140
Arg Asn Glu Lys Tyr Tyr Ile Asp Ile Thr Glu Leu Phe His Glu Val
145 150 155 160
Thr Phe Gln Thr Glu Leu Gly Gln Leu Met Asp Leu Ile Thr Ala Pro
165 170 175
Glu Asp Lys Val Asp Leu Ser Lys Phe Ser Leu Lys Lys His Ser Phe
180 185 190
Ile Val Thr Phe Lys Thr Ala Tyr Tyr Ser Phe Tyr Leu Pro Val Ala
195 200 205
Leu Ala Met Tyr Val Ala Gly Ile Thr Asp Glu Lys Asp Leu Lys Gln
210 215 220
Ala Arg Asp Val Leu Ile Pro Leu Gly Glu Tyr Phe Gln Ile Gln Asp
225 230 235 240
Asp Tyr Leu Asp Cys Phe Gly Thr Pro Glu Gln Ile Gly Lys Ile Gly
245 250 255
Thr Asp Ile Gln Asp Asn Lys Cys Ser Trp Val Ile Asn Lys Ala Leu
260 265 270
Glu Leu Ala Ser Ala Glu Gln Arg Lys Thr Leu Asp Glu Asn Tyr Gly
275 280 285
Lys Lys Asp Ser Val Ala Glu Ala Lys Cys Lys Lys Ile Phe Asn Asp
290 295 300
Leu Lys Ile Glu Gln Leu Tyr His Glu Tyr Glu Glu Ser Ile Ala Lys
305 310 315 320
Asp Leu Lys Ala Lys Ile Ser Gln Val Asp Glu Ser Arg Gly Phe Lys
325 330 335
Ala Asp Val Leu Thr Ala Phe Leu Asn Lys Val Tyr Lys Arg Ser Lys
340 345 350
<210> 11
<211> 1054
<212> PRT
<213> Saccharomyces cerevisiae
<400> 11
Met Pro Pro Leu Phe Lys Gly Leu Lys Gln Met Ala Lys Pro Ile Ala
1 5 10 15
Tyr Val Ser Arg Phe Ser Ala Lys Arg Pro Ile His Ile Ile Leu Phe
20 25 30
Ser Leu Ile Ile Ser Ala Phe Ala Tyr Leu Ser Val Ile Gln Tyr Tyr
35 40 45
Phe Asn Gly Trp Gln Leu Asp Ser Asn Ser Val Phe Glu Thr Ala Pro
50 55 60
Asn Lys Asp Ser Asn Thr Leu Phe Gln Glu Cys Ser His Tyr Tyr Arg
65 70 75 80
Asp Ser Ser Leu Asp Gly Trp Val Ser Ile Thr Ala His Glu Ala Ser
85 90 95
Glu Leu Pro Ala Pro His His Tyr Tyr Leu Leu Asn Leu Asn Phe Asn
100 105 110
Ser Pro Asn Glu Thr Asp Ser Ile Pro Glu Leu Ala Asn Thr Val Phe
115 120 125
Glu Lys Asp Asn Thr Lys Tyr Ile Leu Gln Glu Asp Leu Ser Val Ser
130 135 140
Lys Glu Ile Ser Ser Thr Asp Gly Thr Lys Trp Arg Leu Arg Ser Asp
145 150 155 160
Arg Lys Ser Leu Phe Asp Val Lys Thr Leu Ala Tyr Ser Leu Tyr Asp
165 170 175
Val Phe Ser Glu Asn Val Thr Gln Ala Asp Pro Phe Asp Val Leu Ile
180 185 190
Met Val Thr Ala Tyr Leu Met Met Phe Tyr Thr Ile Phe Gly Leu Phe
195 200 205
Asn Asp Met Arg Lys Thr Gly Ser Asn Phe Trp Leu Ser Ala Ser Thr
210 215 220
Val Val Asn Ser Ala Ser Ser Leu Phe Leu Ala Leu Tyr Val Thr Gln
225 230 235 240
Cys Ile Leu Gly Lys Glu Val Ser Ala Leu Thr Leu Phe Glu Gly Leu
245 250 255
Pro Phe Ile Val Val Val Val Gly Phe Lys His Lys Ile Lys Ile Ala
260 265 270
Gln Tyr Ala Leu Glu Lys Phe Glu Arg Val Gly Leu Ser Lys Arg Ile
275 280 285
Thr Thr Asp Glu Ile Val Phe Glu Ser Val Ser Glu Glu Gly Gly Arg
290 295 300
Leu Ile Gln Asp His Leu Leu Cys Ile Phe Ala Phe Ile Gly Cys Ser
305 310 315 320
Met Tyr Ala His Gln Leu Lys Thr Leu Thr Asn Phe Cys Ile Leu Ser
325 330 335
Ala Phe Ile Leu Ile Phe Glu Leu Ile Leu Thr Pro Thr Phe Tyr Ser
340 345 350
Ala Ile Leu Ala Leu Arg Leu Glu Met Asn Val Ile His Arg Ser Thr
355 360 365
Ile Ile Lys Gln Thr Leu Glu Glu Asp Gly Val Val Pro Ser Thr Ala
370 375 380
Arg Ile Ile Ser Lys Ala Glu Lys Lys Ser Val Ser Ser Phe Leu Asn
385 390 395 400
Leu Ser Val Val Val Ile Ile Met Lys Leu Ser Val Ile Leu Leu Phe
405 410 415
Val Phe Ile Asn Phe Tyr Asn Phe Gly Ala Asn Trp Val Asn Asp Ala
420 425 430
Phe Asn Ser Leu Tyr Phe Asp Lys Glu Arg Val Ser Leu Pro Asp Phe
435 440 445
Ile Thr Ser Asn Ala Ser Glu Asn Phe Lys Glu Gln Ala Ile Val Ser
450 455 460
Val Thr Pro Leu Leu Tyr Tyr Lys Pro Ile Lys Ser Tyr Gln Arg Ile
465 470 475 480
Glu Asp Met Val Leu Leu Leu Leu Arg Asn Val Ser Val Ala Ile Arg
485 490 495
Asp Arg Phe Val Ser Lys Leu Val Leu Ser Ala Leu Val Cys Ser Ala
500 505 510
Val Ile Asn Val Tyr Leu Leu Asn Ala Ala Arg Ile His Thr Ser Tyr
515 520 525
Thr Ala Asp Gln Leu Val Lys Thr Glu Val Thr Lys Lys Ser Phe Thr
530 535 540
Ala Pro Val Gln Lys Ala Ser Thr Pro Val Leu Thr Asn Lys Thr Val
545 550 555 560
Ile Ser Gly Ser Lys Val Lys Ser Leu Ser Ser Ala Gln Ser Ser Ser
565 570 575
Ser Gly Pro Ser Ser Ser Ser Glu Glu Asp Asp Ser Arg Asp Ile Glu
580 585 590
Ser Leu Asp Lys Lys Ile Arg Pro Leu Glu Glu Leu Glu Ala Leu Leu
595 600 605
Ser Ser Gly Asn Thr Lys Gln Leu Lys Asn Lys Glu Val Ala Ala Leu
610 615 620
Val Ile His Gly Lys Leu Pro Leu Tyr Ala Leu Glu Lys Lys Leu Gly
625 630 635 640
Asp Thr Thr Arg Ala Val Ala Val Arg Arg Lys Ala Leu Ser Ile Leu
645 650 655
Ala Glu Ala Pro Val Leu Ala Ser Asp Arg Leu Pro Tyr Lys Asn Tyr
660 665 670
Asp Tyr Asp Arg Val Phe Gly Ala Cys Cys Glu Asn Val Ile Gly Tyr
675 680 685
Met Pro Leu Pro Val Gly Val Ile Gly Pro Leu Val Ile Asp Gly Thr
690 695 700
Ser Tyr His Ile Pro Met Ala Thr Thr Glu Gly Cys Leu Val Ala Ser
705 710 715 720
Ala Met Arg Gly Cys Lys Ala Ile Asn Ala Gly Gly Gly Ala Thr Thr
725 730 735
Val Leu Thr Lys Asp Gly Met Thr Arg Gly Pro Val Val Arg Phe Pro
740 745 750
Thr Leu Lys Arg Ser Gly Ala Cys Lys Ile Trp Leu Asp Ser Glu Glu
755 760 765
Gly Gln Asn Ala Ile Lys Lys Ala Phe Asn Ser Thr Ser Arg Phe Ala
770 775 780
Arg Leu Gln His Ile Gln Thr Cys Leu Ala Gly Asp Leu Leu Phe Met
785 790 795 800
Arg Phe Arg Thr Thr Thr Gly Asp Ala Met Gly Met Asn Met Ile Ser
805 810 815
Lys Gly Val Glu Tyr Ser Leu Lys Gln Met Val Glu Glu Tyr Gly Trp
820 825 830
Glu Asp Met Glu Val Val Ser Val Ser Gly Asn Tyr Cys Thr Asp Lys
835 840 845
Lys Pro Ala Ala Ile Asn Trp Ile Glu Gly Arg Gly Lys Ser Val Val
850 855 860
Ala Glu Ala Thr Ile Pro Gly Asp Val Val Arg Lys Val Leu Lys Ser
865 870 875 880
Asp Val Ser Ala Leu Val Glu Leu Asn Ile Ala Lys Asn Leu Val Gly
885 890 895
Ser Ala Met Ala Gly Ser Val Gly Gly Phe Asn Ala His Ala Ala Asn
900 905 910
Leu Val Thr Ala Val Phe Leu Ala Leu Gly Gln Asp Pro Ala Gln Asn
915 920 925
Val Glu Ser Ser Asn Cys Ile Thr Leu Met Lys Glu Val Asp Gly Asp
930 935 940
Leu Arg Ile Ser Val Ser Met Pro Ser Ile Glu Val Gly Thr Ile Gly
945 950 955 960
Gly Gly Thr Val Leu Glu Pro Gln Gly Ala Met Leu Asp Leu Leu Gly
965 970 975
Val Arg Gly Pro His Ala Thr Ala Pro Gly Thr Asn Ala Arg Gln Leu
980 985 990
Ala Arg Ile Val Ala Cys Ala Val Leu Ala Gly Glu Leu Ser Leu Cys
995 1000 1005
Ala Ala Leu Ala Ala Gly His Leu Val Gln Ser His Met Thr His
1010 1015 1020
Asn Arg Lys Pro Ala Glu Pro Thr Lys Pro Asn Asn Leu Asp Ala
1025 1030 1035
Thr Asp Ile Asn Arg Leu Lys Asp Gly Ser Val Thr Cys Ile Lys
1040 1045 1050
Ser
<210> 12
<211> 288
<212> PRT
<213> Saccharomyces cerevisiae
<400> 12
Met Thr Ala Asp Asn Asn Ser Met Pro His Gly Ala Val Ser Ser Tyr
1 5 10 15
Ala Lys Leu Val Gln Asn Gln Thr Pro Glu Asp Ile Leu Glu Glu Phe
20 25 30
Pro Glu Ile Ile Pro Leu Gln Gln Arg Pro Asn Thr Arg Ser Ser Glu
35 40 45
Thr Ser Asn Asp Glu Ser Gly Glu Thr Cys Phe Ser Gly His Asp Glu
50 55 60
Glu Gln Ile Lys Leu Met Asn Glu Asn Cys Ile Val Leu Asp Trp Asp
65 70 75 80
Asp Asn Ala Ile Gly Ala Gly Thr Lys Lys Val Cys His Leu Met Glu
85 90 95
Asn Ile Glu Lys Gly Leu Leu His Arg Ala Phe Ser Val Phe Ile Phe
100 105 110
Asn Glu Gln Gly Glu Leu Leu Leu Gln Gln Arg Ala Thr Glu Lys Ile
115 120 125
Thr Phe Pro Asp Leu Trp Thr Asn Thr Cys Cys Ser His Pro Leu Cys
130 135 140
Ile Asp Asp Glu Leu Gly Leu Lys Gly Lys Leu Asp Asp Lys Ile Lys
145 150 155 160
Gly Ala Ile Thr Ala Ala Val Arg Lys Leu Asp His Glu Leu Gly Ile
165 170 175
Pro Glu Asp Glu Thr Lys Thr Arg Gly Lys Phe His Phe Leu Asn Arg
180 185 190
Ile His Tyr Met Ala Pro Ser Asn Glu Pro Trp Gly Glu His Glu Ile
195 200 205
Asp Tyr Ile Leu Phe Tyr Lys Ile Asn Ala Lys Glu Asn Leu Thr Val
210 215 220
Asn Pro Asn Val Asn Glu Val Arg Asp Phe Lys Trp Val Ser Pro Asn
225 230 235 240
Asp Leu Lys Thr Met Phe Ala Asp Pro Ser Tyr Lys Phe Thr Pro Trp
245 250 255
Phe Lys Ile Ile Cys Glu Asn Tyr Leu Phe Asn Trp Trp Glu Gln Leu
260 265 270
Asp Asp Leu Ser Glu Val Glu Asn Asp Arg Gln Ile His Arg Met Leu
275 280 285
<210> 13
<211> 396
<212> PRT
<213> Saccharomyces cerevisiae
<400> 13
Met Thr Val Tyr Thr Ala Ser Val Thr Ala Pro Val Asn Ile Ala Thr
1 5 10 15
Leu Lys Tyr Trp Gly Lys Arg Asp Thr Lys Leu Asn Leu Pro Thr Asn
20 25 30
Ser Ser Ile Ser Val Thr Leu Ser Gln Asp Asp Leu Arg Thr Leu Thr
35 40 45
Ser Ala Ala Thr Ala Pro Glu Phe Glu Arg Asp Thr Leu Trp Leu Asn
50 55 60
Gly Glu Pro His Ser Ile Asp Asn Glu Arg Thr Gln Asn Cys Leu Arg
65 70 75 80
Asp Leu Arg Gln Leu Arg Lys Glu Met Glu Ser Lys Asp Ala Ser Leu
85 90 95
Pro Thr Leu Ser Gln Trp Lys Leu His Ile Val Ser Glu Asn Asn Phe
100 105 110
Pro Thr Ala Ala Gly Leu Ala Ser Ser Ala Ala Gly Phe Ala Ala Leu
115 120 125
Val Ser Ala Ile Ala Lys Leu Tyr Gln Leu Pro Gln Ser Thr Ser Glu
130 135 140
Ile Ser Arg Ile Ala Arg Lys Gly Ser Gly Ser Ala Cys Arg Ser Leu
145 150 155 160
Phe Gly Gly Tyr Val Ala Trp Glu Met Gly Lys Ala Glu Asp Gly His
165 170 175
Asp Ser Met Ala Val Gln Ile Ala Asp Ser Ser Asp Trp Pro Gln Met
180 185 190
Lys Ala Cys Val Leu Val Val Ser Asp Ile Lys Lys Asp Val Ser Ser
195 200 205
Thr Gln Gly Met Gln Leu Thr Val Ala Thr Ser Glu Leu Phe Lys Glu
210 215 220
Arg Ile Glu His Val Val Pro Lys Arg Phe Glu Val Met Arg Lys Ala
225 230 235 240
Ile Val Glu Lys Asp Phe Ala Thr Phe Ala Lys Glu Thr Met Met Asp
245 250 255
Ser Asn Ser Phe His Ala Thr Cys Leu Asp Ser Phe Pro Pro Ile Phe
260 265 270
Tyr Met Asn Asp Thr Ser Lys Arg Ile Ile Ser Trp Cys His Thr Ile
275 280 285
Asn Gln Phe Tyr Gly Glu Thr Ile Val Ala Tyr Thr Phe Asp Ala Gly
290 295 300
Pro Asn Ala Val Leu Tyr Tyr Leu Ala Glu Asn Glu Ser Lys Leu Phe
305 310 315 320
Ala Phe Ile Tyr Lys Leu Phe Gly Ser Val Pro Gly Trp Asp Lys Lys
325 330 335
Phe Thr Thr Glu Gln Leu Glu Ala Phe Asn His Gln Phe Glu Ser Ser
340 345 350
Asn Phe Thr Ala Arg Glu Leu Asp Leu Glu Leu Gln Lys Asp Val Ala
355 360 365
Arg Val Ile Leu Thr Gln Val Gly Ser Gly Pro Gln Glu Thr Asn Glu
370 375 380
Ser Leu Ile Asp Ala Lys Thr Gly Leu Pro Lys Glu
385 390 395
<210> 14
<211> 451
<212> PRT
<213> Saccharomyces cerevisiae
<400> 14
Met Ser Glu Leu Arg Ala Phe Ser Ala Pro Gly Lys Ala Leu Leu Ala
1 5 10 15
Gly Gly Tyr Leu Val Leu Asp Thr Lys Tyr Glu Ala Phe Val Val Gly
20 25 30
Leu Ser Ala Arg Met His Ala Val Ala His Pro Tyr Gly Ser Leu Gln
35 40 45
Gly Ser Asp Lys Phe Glu Val Arg Val Lys Ser Lys Gln Phe Lys Asp
50 55 60
Gly Glu Trp Leu Tyr His Ile Ser Pro Lys Ser Gly Phe Ile Pro Val
65 70 75 80
Ser Ile Gly Gly Ser Lys Asn Pro Phe Ile Glu Lys Val Ile Ala Asn
85 90 95
Val Phe Ser Tyr Phe Lys Pro Asn Met Asp Asp Tyr Cys Asn Arg Asn
100 105 110
Leu Phe Val Ile Asp Ile Phe Ser Asp Asp Ala Tyr His Ser Gln Glu
115 120 125
Asp Ser Val Thr Glu His Arg Gly Asn Arg Arg Leu Ser Phe His Ser
130 135 140
His Arg Ile Glu Glu Val Pro Lys Thr Gly Leu Gly Ser Ser Ala Gly
145 150 155 160
Leu Val Thr Val Leu Thr Thr Ala Leu Ala Ser Phe Phe Val Ser Asp
165 170 175
Leu Glu Asn Asn Val Asp Lys Tyr Arg Glu Val Ile His Asn Leu Ala
180 185 190
Gln Val Ala His Cys Gln Ala Gln Gly Lys Ile Gly Ser Gly Phe Asp
195 200 205
Val Ala Ala Ala Ala Tyr Gly Ser Ile Arg Tyr Arg Arg Phe Pro Pro
210 215 220
Ala Leu Ile Ser Asn Leu Pro Asp Ile Gly Ser Ala Thr Tyr Gly Ser
225 230 235 240
Lys Leu Ala His Leu Val Asp Glu Glu Asp Trp Asn Ile Thr Ile Lys
245 250 255
Ser Asn His Leu Pro Ser Gly Leu Thr Leu Trp Met Gly Asp Ile Lys
260 265 270
Asn Gly Ser Glu Thr Val Lys Leu Val Gln Lys Val Lys Asn Trp Tyr
275 280 285
Asp Ser His Met Pro Glu Ser Leu Lys Ile Tyr Thr Glu Leu Asp His
290 295 300
Ala Asn Ser Arg Phe Met Asp Gly Leu Ser Lys Leu Asp Arg Leu His
305 310 315 320
Glu Thr His Asp Asp Tyr Ser Asp Gln Ile Phe Glu Ser Leu Glu Arg
325 330 335
Asn Asp Cys Thr Cys Gln Lys Tyr Pro Glu Ile Thr Glu Val Arg Asp
340 345 350
Ala Val Ala Thr Ile Arg Arg Ser Phe Arg Lys Ile Thr Lys Glu Ser
355 360 365
Gly Ala Asp Ile Glu Pro Pro Val Gln Thr Ser Leu Leu Asp Asp Cys
370 375 380
Gln Thr Leu Lys Gly Val Leu Thr Cys Leu Ile Pro Gly Ala Gly Gly
385 390 395 400
Tyr Asp Ala Ile Ala Val Ile Thr Lys Gln Asp Val Asp Leu Arg Ala
405 410 415
Gln Thr Ala Asn Asp Lys Arg Phe Ser Lys Val Gln Trp Leu Asp Val
420 425 430
Thr Gln Ala Asp Trp Gly Val Arg Lys Glu Lys Asp Pro Glu Thr Tyr
435 440 445
Leu Asp Lys
450
<210> 15
<211> 443
<212> PRT
<213> Saccharomyces cerevisiae
<400> 15
Met Ser Leu Pro Phe Leu Thr Ser Ala Pro Gly Lys Val Ile Ile Phe
1 5 10 15
Gly Glu His Ser Ala Val Tyr Asn Lys Pro Ala Val Ala Ala Ser Val
20 25 30
Ser Ala Leu Arg Thr Tyr Leu Leu Ile Ser Glu Ser Ser Ala Pro Asp
35 40 45
Thr Ile Glu Leu Asp Phe Pro Asp Ile Ser Phe Asn His Lys Trp Ser
50 55 60
Ile Asn Asp Phe Asn Ala Ile Thr Glu Asp Gln Val Asn Ser Gln Lys
65 70 75 80
Leu Ala Lys Ala Gln Gln Ala Thr Asp Gly Leu Ser Gln Glu Leu Val
85 90 95
Ser Leu Leu Asp Pro Leu Leu Ala Gln Leu Ser Glu Ser Phe His Tyr
100 105 110
His Ala Ala Phe Cys Phe Leu Tyr Met Phe Val Cys Leu Cys Pro His
115 120 125
Ala Lys Asn Ile Lys Phe Ser Leu Lys Ser Thr Leu Pro Ile Gly Ala
130 135 140
Gly Leu Gly Ser Ser Ala Ser Ile Ser Val Ser Leu Ala Leu Ala Met
145 150 155 160
Ala Tyr Leu Gly Gly Leu Ile Gly Ser Asn Asp Leu Glu Lys Leu Ser
165 170 175
Glu Asn Asp Lys His Ile Val Asn Gln Trp Ala Phe Ile Gly Glu Lys
180 185 190
Cys Ile His Gly Thr Pro Ser Gly Ile Asp Asn Ala Val Ala Thr Tyr
195 200 205
Gly Asn Ala Leu Leu Phe Glu Lys Asp Ser His Asn Gly Thr Ile Asn
210 215 220
Thr Asn Asn Phe Lys Phe Leu Asp Asp Phe Pro Ala Ile Pro Met Ile
225 230 235 240
Leu Thr Tyr Thr Arg Ile Pro Arg Ser Thr Lys Asp Leu Val Ala Arg
245 250 255
Val Arg Val Leu Val Thr Glu Lys Phe Pro Glu Val Met Lys Pro Ile
260 265 270
Leu Asp Ala Met Gly Glu Cys Ala Leu Gln Gly Leu Glu Ile Met Thr
275 280 285
Lys Leu Ser Lys Cys Lys Gly Thr Asp Asp Glu Ala Val Glu Thr Asn
290 295 300
Asn Glu Leu Tyr Glu Gln Leu Leu Glu Leu Ile Arg Ile Asn His Gly
305 310 315 320
Leu Leu Val Ser Ile Gly Val Ser His Pro Gly Leu Glu Leu Ile Lys
325 330 335
Asn Leu Ser Asp Asp Leu Arg Ile Gly Ser Thr Lys Leu Thr Gly Ala
340 345 350
Gly Gly Gly Gly Cys Ser Leu Thr Leu Leu Arg Arg Asp Ile Thr Gln
355 360 365
Glu Gln Ile Asp Ser Phe Lys Lys Lys Leu Gln Asp Asp Phe Ser Tyr
370 375 380
Glu Thr Phe Glu Thr Asp Leu Gly Gly Thr Gly Cys Cys Leu Leu Ser
385 390 395 400
Ala Lys Asn Leu Asn Lys Asp Leu Lys Ile Lys Ser Leu Val Phe Gln
405 410 415
Leu Phe Glu Asn Lys Thr Thr Thr Lys Gln Gln Ile Asp Asp Leu Leu
420 425 430
Leu Pro Gly Asn Thr Asn Leu Pro Trp Thr Ser
435 440
<210> 16
<211> 491
<212> PRT
<213> Saccharomyces cerevisiae
<400> 16
Met Lys Leu Ser Thr Lys Leu Cys Trp Cys Gly Ile Lys Gly Arg Leu
1 5 10 15
Arg Pro Gln Lys Gln Gln Gln Leu His Asn Thr Asn Leu Gln Met Thr
20 25 30
Glu Leu Lys Lys Gln Lys Thr Ala Glu Gln Lys Thr Arg Pro Gln Asn
35 40 45
Val Gly Ile Lys Gly Ile Gln Ile Tyr Ile Pro Thr Gln Cys Val Asn
50 55 60
Gln Ser Glu Leu Glu Lys Phe Asp Gly Val Ser Gln Gly Lys Tyr Thr
65 70 75 80
Ile Gly Leu Gly Gln Thr Asn Met Ser Phe Val Asn Asp Arg Glu Asp
85 90 95
Ile Tyr Ser Met Ser Leu Thr Val Leu Ser Lys Leu Ile Lys Ser Tyr
100 105 110
Asn Ile Asp Thr Asn Lys Ile Gly Arg Leu Glu Val Gly Thr Glu Thr
115 120 125
Leu Ile Asp Lys Ser Lys Ser Val Lys Ser Val Leu Met Gln Leu Phe
130 135 140
Gly Glu Asn Thr Asp Val Glu Gly Ile Asp Thr Leu Asn Ala Cys Tyr
145 150 155 160
Gly Gly Thr Asn Ala Leu Phe Asn Ser Leu Asn Trp Ile Glu Ser Asn
165 170 175
Ala Trp Asp Gly Arg Asp Ala Ile Val Val Cys Gly Asp Ile Ala Ile
180 185 190
Tyr Asp Lys Gly Ala Ala Arg Pro Thr Gly Gly Ala Gly Thr Val Ala
195 200 205
Met Trp Ile Gly Pro Asp Ala Pro Ile Val Phe Asp Ser Val Arg Ala
210 215 220
Ser Tyr Met Glu His Ala Tyr Asp Phe Tyr Lys Pro Asp Phe Thr Ser
225 230 235 240
Glu Tyr Pro Tyr Val Asp Gly His Phe Ser Leu Thr Cys Tyr Val Lys
245 250 255
Ala Leu Asp Gln Val Tyr Lys Ser Tyr Ser Lys Lys Ala Ile Ser Lys
260 265 270
Gly Leu Val Ser Asp Pro Ala Gly Ser Asp Ala Leu Asn Val Leu Lys
275 280 285
Tyr Phe Asp Tyr Asn Val Phe His Val Pro Thr Cys Lys Leu Val Thr
290 295 300
Lys Ser Tyr Gly Arg Leu Leu Tyr Asn Asp Phe Arg Ala Asn Pro Gln
305 310 315 320
Leu Phe Pro Glu Val Asp Ala Glu Leu Ala Thr Arg Asp Tyr Asp Glu
325 330 335
Ser Leu Thr Asp Lys Asn Ile Glu Lys Thr Phe Val Asn Val Ala Lys
340 345 350
Pro Phe His Lys Glu Arg Val Ala Gln Ser Leu Ile Val Pro Thr Asn
355 360 365
Thr Gly Asn Met Tyr Thr Ala Ser Val Tyr Ala Ala Phe Ala Ser Leu
370 375 380
Leu Asn Tyr Val Gly Ser Asp Asp Leu Gln Gly Lys Arg Val Gly Leu
385 390 395 400
Phe Ser Tyr Gly Ser Gly Leu Ala Ala Ser Leu Tyr Ser Cys Lys Ile
405 410 415
Val Gly Asp Val Gln His Ile Ile Lys Glu Leu Asp Ile Thr Asn Lys
420 425 430
Leu Ala Lys Arg Ile Thr Glu Thr Pro Lys Asp Tyr Glu Ala Ala Ile
435 440 445
Glu Leu Arg Glu Asn Ala His Leu Lys Lys Asn Phe Lys Pro Gln Gly
450 455 460
Ser Ile Glu His Leu Gln Ser Gly Val Tyr Tyr Leu Thr Asn Ile Asp
465 470 475 480
Asp Lys Phe Arg Arg Ser Tyr Asp Val Lys Lys
485 490
<210> 17
<211> 398
<212> PRT
<213> Saccharomyces cerevisiae
<400> 17
Met Ser Gln Asn Val Tyr Ile Val Ser Thr Ala Arg Thr Pro Ile Gly
1 5 10 15
Ser Phe Gln Gly Ser Leu Ser Ser Lys Thr Ala Val Glu Leu Gly Ala
20 25 30
Val Ala Leu Lys Gly Ala Leu Ala Lys Val Pro Glu Leu Asp Ala Ser
35 40 45
Lys Asp Phe Asp Glu Ile Ile Phe Gly Asn Val Leu Ser Ala Asn Leu
50 55 60
Gly Gln Ala Pro Ala Arg Gln Val Ala Leu Ala Ala Gly Leu Ser Asn
65 70 75 80
His Ile Val Ala Ser Thr Val Asn Lys Val Cys Ala Ser Ala Met Lys
85 90 95
Ala Ile Ile Leu Gly Ala Gln Ser Ile Lys Cys Gly Asn Ala Asp Val
100 105 110
Val Val Ala Gly Gly Cys Glu Ser Met Thr Asn Ala Pro Tyr Tyr Met
115 120 125
Pro Ala Ala Arg Ala Gly Ala Lys Phe Gly Gln Thr Val Leu Val Asp
130 135 140
Gly Val Glu Arg Asp Gly Leu Asn Asp Ala Tyr Asp Gly Leu Ala Met
145 150 155 160
Gly Val His Ala Glu Lys Cys Ala Arg Asp Trp Asp Ile Thr Arg Glu
165 170 175
Gln Gln Asp Asn Phe Ala Ile Glu Ser Tyr Gln Lys Ser Gln Lys Ser
180 185 190
Gln Lys Glu Gly Lys Phe Asp Asn Glu Ile Val Pro Val Thr Ile Lys
195 200 205
Gly Phe Arg Gly Lys Pro Asp Thr Gln Val Thr Lys Asp Glu Glu Pro
210 215 220
Ala Arg Leu His Val Glu Lys Leu Arg Ser Ala Arg Thr Val Phe Gln
225 230 235 240
Lys Glu Asn Gly Thr Val Thr Ala Ala Asn Ala Ser Pro Ile Asn Asp
245 250 255
Gly Ala Ala Ala Val Ile Leu Val Ser Glu Lys Val Leu Lys Glu Lys
260 265 270
Asn Leu Lys Pro Leu Ala Ile Ile Lys Gly Trp Gly Glu Ala Ala His
275 280 285
Gln Pro Ala Asp Phe Thr Trp Ala Pro Ser Leu Ala Val Pro Lys Ala
290 295 300
Leu Lys His Ala Gly Ile Glu Asp Ile Asn Ser Val Asp Tyr Phe Glu
305 310 315 320
Phe Asn Glu Ala Phe Ser Val Val Gly Leu Val Asn Thr Lys Ile Leu
325 330 335
Lys Leu Asp Pro Ser Lys Val Asn Val Tyr Gly Gly Ala Val Ala Leu
340 345 350
Gly His Pro Leu Gly Cys Ser Gly Ala Arg Val Val Val Thr Leu Leu
355 360 365
Ser Ile Leu Gln Gln Glu Gly Gly Lys Ile Gly Val Ala Ala Ile Cys
370 375 380
Asn Gly Gly Gly Gly Ala Ser Ser Ile Val Ile Glu Lys Ile
385 390 395
<210> 18
<211> 1045
<212> PRT
<213> Saccharomyces cerevisiae
<400> 18
Met Ser Leu Pro Leu Lys Thr Ile Val His Leu Val Lys Pro Phe Ala
1 5 10 15
Cys Thr Ala Arg Phe Ser Ala Arg Tyr Pro Ile His Val Ile Val Val
20 25 30
Ala Val Leu Leu Ser Ala Ala Ala Tyr Leu Ser Val Thr Gln Ser Tyr
35 40 45
Leu Asn Glu Trp Lys Leu Asp Ser Asn Gln Tyr Ser Thr Tyr Leu Ser
50 55 60
Ile Lys Pro Asp Glu Leu Phe Glu Lys Cys Thr His Tyr Tyr Arg Ser
65 70 75 80
Pro Val Ser Asp Thr Trp Lys Leu Leu Ser Ser Lys Glu Ala Ala Asp
85 90 95
Ile Tyr Thr Pro Phe His Tyr Tyr Leu Ser Thr Ile Ser Phe Gln Ser
100 105 110
Lys Asp Asn Ser Thr Thr Leu Pro Ser Leu Asp Asp Val Ile Tyr Ser
115 120 125
Val Asp His Thr Arg Tyr Leu Leu Ser Glu Glu Pro Lys Ile Pro Thr
130 135 140
Glu Leu Val Ser Glu Asn Gly Thr Lys Trp Arg Leu Arg Asn Asn Ser
145 150 155 160
Asn Phe Ile Leu Asp Leu His Asn Ile Tyr Arg Asn Met Val Lys Gln
165 170 175
Phe Ser Asn Lys Thr Ser Glu Phe Asp Gln Phe Asp Leu Phe Ile Ile
180 185 190
Leu Ala Ala Tyr Leu Thr Leu Phe Tyr Thr Leu Cys Cys Leu Phe Asn
195 200 205
Asp Met Arg Lys Ile Gly Ser Lys Phe Trp Leu Ser Phe Ser Ala Leu
210 215 220
Ser Asn Ser Ala Cys Ala Leu Tyr Leu Ser Leu Tyr Thr Thr His Ser
225 230 235 240
Leu Leu Lys Lys Pro Ala Ser Leu Leu Ser Leu Val Ile Gly Leu Pro
245 250 255
Phe Ile Val Val Ile Ile Gly Phe Lys His Lys Val Arg Leu Ala Ala
260 265 270
Phe Ser Leu Gln Lys Phe His Arg Ile Ser Ile Asp Lys Lys Ile Thr
275 280 285
Val Ser Asn Ile Ile Tyr Glu Ala Met Phe Gln Glu Gly Ala Tyr Leu
290 295 300
Ile Arg Asp Tyr Leu Phe Tyr Ile Ser Ser Phe Ile Gly Cys Ala Ile
305 310 315 320
Tyr Ala Arg His Leu Pro Gly Leu Val Asn Phe Cys Ile Leu Ser Thr
325 330 335
Phe Met Leu Val Phe Asp Leu Leu Leu Ser Ala Thr Phe Tyr Ser Ala
340 345 350
Ile Leu Ser Met Lys Leu Glu Ile Asn Ile Ile His Arg Ser Thr Val
355 360 365
Ile Arg Gln Thr Leu Glu Glu Asp Gly Val Val Pro Thr Thr Ala Asp
370 375 380
Ile Ile Tyr Lys Asp Glu Thr Ala Ser Glu Pro His Phe Leu Arg Ser
385 390 395 400
Asn Val Ala Ile Ile Leu Gly Lys Ala Ser Val Ile Gly Leu Leu Leu
405 410 415
Leu Ile Asn Leu Tyr Val Phe Thr Asp Lys Leu Asn Ala Thr Ile Leu
420 425 430
Asn Thr Val Tyr Phe Asp Ser Thr Ile Tyr Ser Leu Pro Asn Phe Ile
435 440 445
Asn Tyr Lys Asp Ile Gly Asn Leu Ser Asn Gln Val Ile Ile Ser Val
450 455 460
Leu Pro Lys Gln Tyr Tyr Thr Pro Leu Lys Lys Tyr His Gln Ile Glu
465 470 475 480
Asp Ser Val Leu Leu Ile Ile Asp Ser Val Ser Asn Ala Ile Arg Asp
485 490 495
Gln Phe Ile Ser Lys Leu Leu Phe Phe Ala Phe Ala Val Ser Ile Ser
500 505 510
Ile Asn Val Tyr Leu Leu Asn Ala Ala Lys Ile His Thr Gly Tyr Met
515 520 525
Asn Phe Gln Pro Gln Ser Asn Lys Ile Asp Asp Leu Val Val Gln Gln
530 535 540
Lys Ser Ala Thr Ile Glu Phe Ser Glu Thr Arg Ser Met Pro Ala Ser
545 550 555 560
Ser Gly Leu Glu Thr Pro Val Thr Ala Lys Asp Ile Ile Ile Ser Glu
565 570 575
Glu Ile Gln Asn Asn Glu Cys Val Tyr Ala Leu Ser Ser Gln Asp Glu
580 585 590
Pro Ile Arg Pro Leu Ser Asn Leu Val Glu Leu Met Glu Lys Glu Gln
595 600 605
Leu Lys Asn Met Asn Asn Thr Glu Val Ser Asn Leu Val Val Asn Gly
610 615 620
Lys Leu Pro Leu Tyr Ser Leu Glu Lys Lys Leu Glu Asp Thr Thr Arg
625 630 635 640
Ala Val Leu Val Arg Arg Lys Ala Leu Ser Thr Leu Ala Glu Ser Pro
645 650 655
Ile Leu Val Ser Glu Lys Leu Pro Phe Arg Asn Tyr Asp Tyr Asp Arg
660 665 670
Val Phe Gly Ala Cys Cys Glu Asn Val Ile Gly Tyr Met Pro Ile Pro
675 680 685
Val Gly Val Ile Gly Pro Leu Ile Ile Asp Gly Thr Ser Tyr His Ile
690 695 700
Pro Met Ala Thr Thr Glu Gly Cys Leu Val Ala Ser Ala Met Arg Gly
705 710 715 720
Cys Lys Ala Ile Asn Ala Gly Gly Gly Ala Thr Thr Val Leu Thr Lys
725 730 735
Asp Gly Met Thr Arg Gly Pro Val Val Arg Phe Pro Thr Leu Ile Arg
740 745 750
Ser Gly Ala Cys Lys Ile Trp Leu Asp Ser Glu Glu Gly Gln Asn Ser
755 760 765
Ile Lys Lys Ala Phe Asn Ser Thr Ser Arg Phe Ala Arg Leu Gln His
770 775 780
Ile Gln Thr Cys Leu Ala Gly Asp Leu Leu Phe Met Arg Phe Arg Thr
785 790 795 800
Thr Thr Gly Asp Ala Met Gly Met Asn Met Ile Ser Lys Gly Val Glu
805 810 815
Tyr Ser Leu Lys Gln Met Val Glu Glu Tyr Gly Trp Glu Asp Met Glu
820 825 830
Val Val Ser Val Ser Gly Asn Tyr Cys Thr Asp Lys Lys Pro Ala Ala
835 840 845
Ile Asn Trp Ile Glu Gly Arg Gly Lys Ser Val Val Ala Glu Ala Thr
850 855 860
Ile Pro Gly Asp Val Val Lys Ser Val Leu Lys Ser Asp Val Ser Ala
865 870 875 880
Leu Val Glu Leu Asn Ile Ser Lys Asn Leu Val Gly Ser Ala Met Ala
885 890 895
Gly Ser Val Gly Gly Phe Asn Ala His Ala Ala Asn Leu Val Thr Ala
900 905 910
Leu Phe Leu Ala Leu Gly Gln Asp Pro Ala Gln Asn Val Glu Ser Ser
915 920 925
Asn Cys Ile Thr Leu Met Lys Glu Val Asp Gly Asp Leu Arg Ile Ser
930 935 940
Val Ser Met Pro Ser Ile Glu Val Gly Thr Ile Gly Gly Gly Thr Val
945 950 955 960
Leu Glu Pro Gln Gly Ala Met Leu Asp Leu Leu Gly Val Arg Gly Pro
965 970 975
His Pro Thr Glu Pro Gly Ala Asn Ala Arg Gln Leu Ala Arg Ile Ile
980 985 990
Ala Cys Ala Val Leu Ala Gly Glu Leu Ser Leu Cys Ser Ala Leu Ala
995 1000 1005
Ala Gly His Leu Val Gln Ser His Met Thr His Asn Arg Lys Thr
1010 1015 1020
Asn Lys Ala Asn Glu Leu Pro Gln Pro Ser Asn Lys Gly Pro Pro
1025 1030 1035
Cys Lys Thr Ser Ala Leu Leu
1040 1045
<210> 19
<211> 479
<212> PRT
<213> Aspergillus japonicus
<400> 19
Met Thr Ala Gly Gln Gly Ile Lys Thr Gly Asn Ala Ser Asp Cys Glu
1 5 10 15
Val Tyr Arg Thr Leu Ser Val Ala Leu Asp Phe Ala Asn Gln Asp Glu
20 25 30
Glu Leu Trp Trp His Ser Thr Ala Pro Met Phe Ala Gln Met Leu Gln
35 40 45
Ser Thr Asn Tyr Asn Leu His Ala Gln Tyr Lys His Leu Leu Ile Tyr
50 55 60
Lys Lys Asn Val Ile Pro Phe Leu Gly Val Tyr Pro Thr Asn Asp Lys
65 70 75 80
Pro Arg Trp Leu Ser Ile Leu Thr Arg Tyr Gly Thr Pro Phe Glu Leu
85 90 95
Ser Leu Asn Cys Ser Gly Pro Leu Val Arg Tyr Thr Tyr Glu Pro Ile
100 105 110
Asn Ala Ala Thr Gly Thr Ala Arg Asp Pro Phe Asn Thr His Ala Val
115 120 125
Trp Asp Ser Leu Glu Gln Leu Met Ala Leu Gln Ser Gly Ile Asp Leu
130 135 140
Asp Leu Phe Arg His Phe Lys Asn Asp Leu Thr Leu Ser Ala Glu Glu
145 150 155 160
Ser Glu Tyr Leu Tyr Lys Asn Asn Leu Val Gly Glu Gln Ile Arg Thr
165 170 175
Gln Asn Lys Leu Ala Leu Asp Leu Gln Asp Gly Glu Phe Val Val Lys
180 185 190
Thr Tyr Ile Tyr Pro Ala Leu Lys Ser Leu Ala Thr Gly Arg Ser Ile
195 200 205
His Glu Leu Val Phe Gly Ser Ala Phe Arg Trp Ser Lys Gln Tyr Pro
210 215 220
Glu Leu Arg Lys Pro Leu Asp Thr Leu Glu Gln Tyr Val Tyr Ser Arg
225 230 235 240
Gly Pro Ser Ser Thr Ala Ser Pro Arg Leu Leu Ser Cys Asp Leu Ile
245 250 255
Asp Pro Thr Lys Ser Arg Ile Lys Ile Tyr Leu Leu Glu Arg Met Val
260 265 270
Thr Leu Glu Ala Leu Glu Asp Leu Trp Thr Met Gly Gly Glu Arg Thr
275 280 285
Asp Ala Ser Thr Leu Ala Gly Leu Glu Met Ile Arg Glu Leu Trp Glu
290 295 300
Leu Ile Arg Leu Pro Ala Gly Leu Gln Ser Tyr Pro Ala Pro Tyr Leu
305 310 315 320
Pro Ile Gly Thr Ile Pro Asp Glu Gln Leu Pro Leu Met Ala Asn Tyr
325 330 335
Thr Ile His His Asp Asp Pro Val Pro Glu Pro Gln Val Tyr Phe Thr
340 345 350
Thr Phe Gly Arg Asn Asp Met Gln Ile Ala Asp Ala Leu Ala Thr Phe
355 360 365
Phe Glu Arg Arg Gly Trp His Glu Met Ala Arg Gln Tyr Lys Ala Glu
370 375 380
Leu Cys Ser His Tyr Pro His Ala Asp His Glu Thr Leu Asn Tyr Leu
385 390 395 400
His Ala Tyr Ile Ser Phe Ser Tyr Arg Lys Asn Lys Pro Tyr Leu Ser
405 410 415
Val Tyr Leu Gln Ser Leu Glu Thr Gly Asp Trp Val Thr Ser Ser Phe
420 425 430
Asn Ser Val His Val Asp Pro Gly Leu Ser Ala Thr Val Gln Glu Leu
435 440 445
Ser Lys Leu Thr Lys Thr Ala Gly Thr Thr Val Arg Glu Thr Lys Leu
450 455 460
Pro Leu Thr Pro Asp Gly Ser Glu Pro Gly Val Ile Thr Gln Tyr
465 470 475
<210> 20
<211> 441
<212> PRT
<213> Paecilomyces divaricatus
<400> 20
Met Ser Ser Arg Lys Thr Arg Asn Ala Ser Ser Ser Glu Val Tyr Gln
1 5 10 15
Thr Leu Ser Ser Leu Phe Asp Phe Pro Asp Glu Glu Gln Lys Leu Trp
20 25 30
Trp His Ser Thr Ala Pro Met Phe Ala Asp Met Leu Gln Thr Ala Asn
35 40 45
Tyr Asp Thr His Ala Gln Tyr Arg His Leu Gly Ile Tyr Lys Lys Tyr
50 55 60
Val Ile Pro Phe Leu Gly Val Tyr Pro Thr Asn Asp Arg Asp Arg Trp
65 70 75 80
Leu Ser Ile Leu Thr Arg Tyr Gly Thr Pro Phe Glu Leu Ser Leu Asn
85 90 95
Cys Ser Asp Asp Val Val Arg Tyr Thr Phe Glu Pro Ile Asn Ala Ala
100 105 110
Thr Gly Thr Ile Gln Asp Pro Phe Asn Thr His Ala Ile Trp Asp Cys
115 120 125
Leu Gln Asn Phe Ser Pro Met Leu Lys Gly Val Asp Leu Glu Trp Phe
130 135 140
Ser Tyr Phe Lys Glu Lys Leu Thr Leu Asn Ala Asp Glu Ser Ala Phe
145 150 155 160
Leu Val Glu Asn Asn Leu Ala Asp Asp Pro Ile Lys Thr Gln Asn Lys
165 170 175
Leu Ala Leu Asp Leu Lys Asn Ser Gly Phe Val Leu Lys Thr Tyr Ile
180 185 190
Tyr Pro Ala Leu Lys Ser Ile Ala Thr Gly Leu Ala Ala Pro Leu Ser
195 200 205
Val Ile Asp Glu Tyr Val His Ser Arg Gly Pro Asn Gly Thr Ala Gly
210 215 220
Pro Arg Leu Phe Ser Cys Asp Leu Val His Pro Ser Lys Ser Arg Ile
225 230 235 240
Lys Ile Tyr Ile Leu Glu Arg Met Val Ser Leu Ala Ala Met Glu Asp
245 250 255
Leu Trp Thr Leu Gly Gly Arg Arg Lys Asp His Ser Thr Leu Ala Gly
260 265 270
Leu Asn Met Ile Arg Glu Leu Trp Glu Leu Ile Gln Leu Pro Ala Gly
275 280 285
Leu Arg Ser Tyr Pro Glu Pro Tyr Leu Pro Leu Gly Thr Ile Pro Asn
290 295 300
Glu Gln Leu Pro Leu Met Ala Asn Tyr Thr Leu His Gln Asp Asp Pro
305 310 315 320
Trp Pro Glu Pro Gln Val Tyr Phe Thr Pro Phe Gly Met Asn Asp Met
325 330 335
Ala Ile Ala Asp Ala Leu Thr Thr Phe Met Glu Arg Arg Gly Trp Asn
340 345 350
Asp Met Ala Gln Ser Tyr Lys Asp Asn Leu Cys Ser Tyr Tyr Pro Asn
355 360 365
Ala Asp His Asp His Leu Asn Tyr Val His Ala Tyr Ile Ser Phe Ser
370 375 380
Tyr Arg Asn Asp Lys Pro Tyr Leu Ser Val Tyr Leu His Ser Phe Glu
385 390 395 400
Thr Gly Asp Trp Lys Ile Ser Asn Phe Thr Asn Pro Glu Ser Lys Cys
405 410 415
Lys Glu Pro Gln Arg Val Ser Val Arg Phe Pro Val Gln Gly Arg Gly
420 425 430
Arg Gly Arg Ile Pro Ala Ser Gly Tyr
435 440
<210> 21
<211> 457
<212> PRT
<213> Trichophyton equinum
<400> 21
Met Gly Ser Ile Glu Ile Pro Asn Cys Ser Gly Ser Ile Val Tyr Lys
1 5 10 15
Thr Ile Ser Asp Phe Ile Asp Phe Pro Ser His Glu Gln Lys Leu Trp
20 25 30
Trp His Ser Thr Ala Pro Met Phe Ala Glu Met Leu Arg Val Ala Gly
35 40 45
Tyr Asp Leu His Ser Gln Tyr Lys Ile Leu Gly Ile Phe Leu Asn His
50 55 60
Val Ile Pro Phe Leu Gly Val Tyr Pro Thr Arg Ile Asn Asn Arg Trp
65 70 75 80
Leu Ser Ile Leu Thr Arg Tyr Gly Thr Pro Phe Glu Leu Ser Leu Asn
85 90 95
Cys Ser Gln Ser Leu Val Arg Tyr Thr Tyr Glu Pro Ile Asn Ser Ala
100 105 110
Thr Gly Thr Val Lys Asp Pro Phe Asn Thr His Ser Ile Trp Asp Ala
115 120 125
Leu Asp Arg Leu Met Pro Leu Gln Lys Gly Ile Asp Leu Glu Phe Phe
130 135 140
Lys His Leu Lys Gln Asp Leu Thr Val Asp Asp Gln Asp Ser Ala Tyr
145 150 155 160
Leu Leu Glu Asn Asn Leu Val Gly Gly Gln Ile Arg Thr Gln Asn Lys
165 170 175
Leu Ala Leu Asp Leu Lys Gly Gly Asn Phe Val Leu Lys Thr Tyr Ile
180 185 190
Tyr Pro Ala Leu Lys Ala Leu Ala Thr Gly Lys Ser Ile Lys Thr Leu
195 200 205
Met Phe Asp Ser Val Tyr Arg Leu Cys Arg Gln Asn Pro Ser Leu Glu
210 215 220
Ala Pro Leu Arg Ala Leu Glu Glu Tyr Val Asp Ser Lys Gly Pro Asn
225 230 235 240
Ser Thr Ser Ser Pro Arg Leu Leu Ser Cys Asp Leu Ile Asp Pro Ser
245 250 255
Lys Ser Arg Val Lys Ile Tyr Ile Leu Glu Leu Asn Val Thr Leu Glu
260 265 270
Ala Met Glu Asp Leu Trp Thr Met Gly Gly Arg Leu Asn Asp Ala Ser
275 280 285
Thr Leu Ala Gly Leu Glu Met Leu Arg Glu Leu Trp Asp Leu Ile Lys
290 295 300
Leu Pro Pro Gly Met Arg Glu Tyr Pro Glu Pro Phe Leu Gln Leu Gly
305 310 315 320
Thr Ile Pro Asp Glu Gln Leu Pro Leu Met Ala Asn Tyr Thr Leu His
325 330 335
His Asp Gln Ala Met Pro Glu Pro Gln Val Tyr Phe Thr Thr Phe Gly
340 345 350
Leu Asn Asp Gly Arg Ile Ala Asp Gly Leu Val Thr Phe Phe Glu Arg
355 360 365
Arg Gly Trp Asn His Met Ala Gln Thr Tyr Lys Asp Ser Leu Arg Ala
370 375 380
Tyr Tyr Pro His Ala Asp Gln Glu Thr Leu Asn Tyr Leu His Ala Tyr
385 390 395 400
Ile Ser Phe Ser Tyr Arg Lys Gly Lys Pro Tyr Leu Ser Val Tyr Leu
405 410 415
Gln Thr Phe Glu Thr Gly Asp Trp Pro Ile Cys Lys Cys Ala Ala Val
420 425 430
Glu Arg Arg Lys Trp Asn Ile Asn Leu Thr Asp Ile Val Ser Gln Leu
435 440 445
Trp Asp Pro Gly Gly Gln Ala Ser Pro
450 455
<210> 22
<211> 459
<212> PRT
<213> Aspergillus fumigatus
<400> 22
Met Lys Ala Ala Asn Ala Ser Ser Ala Glu Ala Tyr Arg Val Leu Ser
1 5 10 15
Arg Ala Phe Arg Phe Asp Asn Glu Asp Gln Lys Leu Trp Trp His Ser
20 25 30
Thr Ala Pro Met Phe Ala Lys Met Leu Glu Thr Ala Asn Tyr Thr Thr
35 40 45
Pro Cys Gln Tyr Gln Tyr Leu Ile Thr Tyr Lys Glu Cys Val Ile Pro
50 55 60
Ser Leu Gly Cys Tyr Pro Thr Asn Ser Ala Pro Arg Trp Leu Ser Ile
65 70 75 80
Leu Thr Arg Tyr Gly Thr Pro Phe Glu Leu Ser Leu Asn Cys Ser Asn
85 90 95
Ser Ile Val Arg Tyr Thr Phe Glu Pro Ile Asn Gln His Thr Gly Thr
100 105 110
Asp Lys Asp Pro Phe Asn Thr His Ala Ile Trp Glu Ser Leu Gln His
115 120 125
Leu Leu Pro Leu Glu Lys Ser Ile Asp Leu Glu Trp Phe Arg His Phe
130 135 140
Lys His Asp Leu Thr Leu Asn Ser Glu Glu Ser Ala Phe Leu Ala His
145 150 155 160
Asn Asp Arg Leu Val Gly Gly Thr Ile Arg Thr Gln Asn Lys Leu Ala
165 170 175
Leu Asp Leu Lys Asp Gly Arg Phe Ala Leu Lys Thr Tyr Ile Tyr Pro
180 185 190
Ala Leu Lys Ala Val Val Thr Gly Lys Thr Ile His Glu Leu Val Phe
195 200 205
Gly Ser Val Arg Arg Leu Ala Val Arg Glu Pro Arg Ile Leu Pro Pro
210 215 220
Leu Asn Met Leu Glu Glu Tyr Ile Arg Ser Arg Gly Ser Lys Ser Thr
225 230 235 240
Ala Ser Pro Arg Leu Val Ser Cys Asp Leu Thr Ser Pro Ala Lys Ser
245 250 255
Arg Ile Lys Ile Tyr Leu Leu Glu Gln Met Val Ser Leu Glu Ala Met
260 265 270
Glu Asp Leu Trp Thr Leu Gly Gly Arg Arg Arg Asp Ala Ser Thr Leu
275 280 285
Glu Gly Leu Ser Leu Val Arg Glu Leu Trp Asp Leu Ile Gln Leu Ser
290 295 300
Pro Gly Leu Lys Ser Tyr Pro Ala Pro Tyr Leu Pro Leu Gly Val Ile
305 310 315 320
Pro Asp Glu Arg Leu Pro Leu Met Ala Asn Phe Thr Leu His Gln Asn
325 330 335
Asp Pro Val Pro Glu Pro Gln Val Tyr Phe Thr Thr Phe Gly Met Asn
340 345 350
Asp Met Ala Val Ala Asp Ala Leu Thr Thr Phe Phe Glu Arg Arg Gly
355 360 365
Trp Ser Glu Met Ala Arg Thr Tyr Glu Thr Thr Leu Lys Ser Tyr Tyr
370 375 380
Pro His Ala Asp His Asp Lys Leu Asn Tyr Leu His Ala Tyr Ile Ser
385 390 395 400
Phe Ser Tyr Arg Asp Arg Thr Pro Tyr Leu Ser Val Tyr Leu Gln Ser
405 410 415
Phe Glu Thr Gly Asp Trp Ala Val Ala Asn Leu Ser Glu Ser Lys Val
420 425 430
Lys Cys Gln Asp Ala Ala Cys Gln Pro Thr Ser Leu Pro Pro Asp Leu
435 440 445
Ser Lys Thr Gly Val Tyr Tyr Ser Gly Leu His
450 455
<210> 23
<211> 456
<212> PRT
<213> Epichloe brachyelytri
<400> 23
Met Ala Met Ala Lys Thr Leu His Gln Glu Val Tyr Gln Thr Leu Ser
1 5 10 15
Glu Ile Phe Asp Phe Ala Asn Asn Asp Gln Arg Leu Trp Trp His Ser
20 25 30
Thr Ala Pro Met Phe Glu Lys Met Leu Gln Thr Ala Asn Tyr Ser Ile
35 40 45
His Ala Gln Tyr Arg His Leu Gly Ile Tyr Lys Arg His Ile Ile Pro
50 55 60
Phe Leu Gly Val Tyr Pro Thr Arg Ser Gly Gln Arg Trp Leu Ser Ile
65 70 75 80
Leu Thr Arg Tyr Gly Thr Pro Phe Glu Leu Ser Leu Asn Cys Ser Asp
85 90 95
Ser Ile Val Arg Tyr Thr Tyr Glu Pro Ile Asn Ala Ala Thr Gly Ser
100 105 110
His Leu Asp Pro Phe Asn Thr Phe Ala Ile Trp Glu Ala Leu Lys Asn
115 120 125
Leu Met Asp Phe Gln Leu Gly Ile Asp Leu Glu Trp Phe Ser Tyr Phe
130 135 140
Lys Gln Glu Leu Thr Leu Asp Ala Asn Glu Ser Thr Tyr Leu His Ser
145 150 155 160
Gln Asn Leu Val Lys Glu Gln Ile Lys Thr Gln Asn Lys Leu Ala Leu
165 170 175
Asp Leu Lys Gly Asp Lys Phe Val Leu Lys Thr Tyr Ile Tyr Pro Glu
180 185 190
Leu Lys Ser Val Ala Thr Gly Lys Ser Val Gln Glu Leu Val Phe Gly
195 200 205
Ser Val Arg Lys Leu Ala Gln Lys His Lys Ser Ile Arg Pro Ala Phe
210 215 220
Glu Met Leu Glu Asp Tyr Val Gln Ser Arg Asn Glu Val Ser Thr Thr
225 230 235 240
Asp Gly Ser His Ser Thr Pro Leu Ser Ser Arg Leu Leu Ser Cys Asp
245 250 255
Leu Met Ser Pro Thr Lys Ser Arg Val Lys Ile Tyr Leu Leu Glu Arg
260 265 270
Met Val Ser Leu Pro Ala Ile Glu Asp Leu Trp Thr Leu Gly Gly Arg
275 280 285
Arg Glu Asp Gln Ser Thr Ile Glu Gly Leu Glu Met Ile Arg Glu Leu
290 295 300
Trp Gly Leu Leu Asn Met Ser Pro Gly Leu Arg Ala Tyr Pro Lys Pro
305 310 315 320
Tyr Leu Pro Leu Gly Ala Ile Pro Asn Glu Gln Leu Pro Ser Met Ala
325 330 335
Asn Tyr Thr Leu His His Asn Asp Pro Met Pro Glu Pro Gln Val Tyr
340 345 350
Phe Thr Val Phe Gly Met Asn Asp Met Glu Val Thr Asp Ala Leu Thr
355 360 365
Thr Phe Phe Met Arg His Asp Trp Ser Asp Met Ala Ser Lys Tyr Lys
370 375 380
Ala Ser Leu Arg Glu Ser Phe Pro His His Asn Tyr Glu Ala Leu Asn
385 390 395 400
Tyr Ile His Ser Tyr Ile Ser Phe Ser Tyr Arg Lys Asn Lys Pro Tyr
405 410 415
Leu Ser Val Tyr Leu His Ser Phe Glu Thr Gly Asp Trp Pro Val Phe
420 425 430
Pro Asp Ser Leu Thr Ala Phe Asp Ala Tyr Arg Arg Asp Ser Thr Cys
435 440 445
Tyr Lys Lys Ile Trp Leu Trp Gln
450 455
<210> 24
<211> 455
<212> PRT
<213> Metarhizium robertsii
<400> 24
Met Lys Ile Glu Glu Arg Met Gly His Ser Val Tyr Glu Thr Leu Ser
1 5 10 15
Leu Ile Phe Asp Phe Pro Asn Asp Asp Gln Lys Leu Trp Trp His Ser
20 25 30
Thr Ala Pro Met Phe Glu Ala Met Leu Gln Thr Ala Gly His Thr Ile
35 40 45
Asp Thr Gln Tyr Arg His Leu Gly Ile Tyr Lys Lys His Val Ile Pro
50 55 60
Phe Leu Gly Val Tyr Pro Thr Asn Asp Lys Glu Arg Trp Leu Ser Ile
65 70 75 80
Leu Thr Arg Tyr Gly Ile Pro Phe Glu Leu Ser Leu Asn Cys Ser Asn
85 90 95
Ser Val Val Arg Tyr Thr Tyr Glu Pro Ile Asn Ala Ala Ser Gly Thr
100 105 110
Gln Lys Asp Pro Tyr Asn Thr Leu Ala Ile Trp Glu Ser Leu Gln Lys
115 120 125
Leu Val Gln Ile Gln Ala Gly Ile Asp Val Glu Trp Phe Lys Tyr Phe
130 135 140
Lys Arg Glu Leu Thr Leu Asn Thr Ala Glu Ser Ala Tyr Leu Phe Asp
145 150 155 160
Asn Gly Leu Ala Lys Asp Glu Ile Lys Thr Gln Asn Lys Leu Ala Leu
165 170 175
Asp Leu Lys Gly Asn Gln Phe Ala Val Lys Val Tyr Ile Tyr Pro Ala
180 185 190
Leu Lys Ser Leu Ala Thr Gly Lys Ser Met His Glu Leu Ile Phe Gly
195 200 205
Ser Val His Lys Leu Ser Leu Gln Tyr Ser Ser Ile Arg Ala Ala Phe
210 215 220
Glu Arg Leu Glu Gly Phe Val Ile Ser Arg Asn Ser Arg Ala Gln Thr
225 230 235 240
Gly Asp Cys Gly Ala Leu His Pro Arg Leu Leu Ser Cys Asp Leu Val
245 250 255
Asp Pro Ala Lys Ser Arg Ile Lys Ile Tyr Leu Leu Glu Gln Met Val
260 265 270
Ser Leu Ser Ala Met Glu Asp Leu Trp Thr Leu Gly Gly Gln Leu Thr
275 280 285
Asp Gln Ser Thr Met Asp Gly Leu Asp Leu Ile Arg Glu Leu Trp Asp
290 295 300
Leu Leu Arg Ile Pro Pro Gly Leu Arg Ser Tyr Pro Glu Pro Tyr Leu
305 310 315 320
Ala Leu Gly Lys Val Pro Asp Glu Arg Leu Pro Ser Met Val Asn Tyr
325 330 335
Thr Leu His His Asp Asp Pro Thr Pro Glu Pro Gln Val Tyr Phe Thr
340 345 350
Val Phe Gly Met Ser Asp Leu Asp Val Thr Asn Ala Leu Thr Val Phe
355 360 365
Phe Glu Arg Arg Gly Trp Ser Glu Met Ala Thr Lys Tyr Arg Ala Phe
370 375 380
Leu Gln Lys Ser Phe Pro His Asp Asp His Glu Ser Leu Asn Tyr Ile
385 390 395 400
His Thr Tyr Ile Ser Phe Ser Tyr Arg Asn Asn Lys Pro Tyr Leu Ser
405 410 415
Val Tyr Leu His Ser Phe Glu Ser Gly Asn Trp Pro Val Leu Pro Asp
420 425 430
Val Pro Val Ala Phe Asp Ala Phe Arg Arg Glu Pro Ser Pro Tyr Lys
435 440 445
Lys Ile Glu Leu Gly Leu Asp
450 455
<210> 25
<211> 456
<212> PRT
<213> Periglandula ipomoeae
<400> 25
Met Ala Thr Val Asn Glu Ser Gly Arg Gly Val Tyr Asp Thr Leu Ser
1 5 10 15
Leu Ile Phe Asp Phe Pro Asp Asp Glu Gln Arg Leu Trp Trp His Ser
20 25 30
Thr Ala Pro Met Phe Ala Ala Met Leu Gln Thr Ala Gly His Asn Val
35 40 45
His Asp Gln Tyr Arg His Leu Gly Ile Tyr Lys Lys Asn Ile Ile Pro
50 55 60
Phe Leu Gly Val Tyr Pro Thr Glu Gly Lys Gly Arg Trp Leu Ser Ile
65 70 75 80
Leu Thr Arg Tyr Gly Ile Pro Phe Glu Leu Ser Leu Asn Cys Ser Asn
85 90 95
Ser Ile Val Arg Tyr Thr Tyr Glu Pro Ile Asn Glu Ala Thr Gly Thr
100 105 110
Asp Lys Asp Pro Tyr Asn Thr Leu Ala Ile Leu Glu Ser Leu Gln Lys
115 120 125
Leu Val Gln Ile Gln Ser Gly Ile Asp Leu Glu Trp Phe Asn Tyr Phe
130 135 140
Lys His Glu Leu Thr Leu Asn Gly Thr Glu Ser Ala Tyr Leu Arg Ser
145 150 155 160
Asn Asp Leu Val Asn Cys Gln Ile Lys Thr Gln Asn Lys Leu Ala Leu
165 170 175
Asp Leu Lys Gly Asn Gln Phe Ala Leu Lys Val Tyr Ile Tyr Pro Glu
180 185 190
Leu Lys Ser Thr Ala Thr Gly Lys Ser Ile His Glu Leu Ile Phe Gly
195 200 205
Ser Val Arg Lys Leu Ser Leu Glu His Pro Ser Ile Gln Pro Ala Phe
210 215 220
Gln Val Leu Asp Asp Tyr Val Ala Ser Arg Asn Ile Ser Ala Glu Thr
225 230 235 240
Gly Gly Glu Tyr Ser Ala Leu Gln Pro Arg Leu Leu Ser Cys Asp Leu
245 250 255
Ile Asn Pro Ala Lys Ser Arg Val Lys Ile Tyr Leu Leu Glu Arg Thr
260 265 270
Val Ser Leu Ser Ala Met Glu Asp Leu Trp Thr Leu Gly Gly Arg Arg
275 280 285
Thr Asp Pro Ser Thr Met Asp Gly Leu Asp Met Val Arg Glu Leu Trp
290 295 300
Asn Leu Leu Glu Met Pro Ala Gly Leu Gln Ala Tyr Pro Lys Pro Tyr
305 310 315 320
Leu Gln Leu Gly Lys Ile Pro Asn Glu Gln Leu Pro Ser Met Ala Asn
325 330 335
Tyr Thr Leu His His Asn Asp Pro Met Pro Glu Pro Gln Val Tyr Phe
340 345 350
Thr Val Phe Gly Met Ser Asp Phe Lys Val Thr Asn Ala Leu Thr Val
355 360 365
Phe Phe Glu Arg Arg Gly Trp Asn Glu Met Ala Arg Lys Tyr Arg Val
370 375 380
Phe Leu Gln Glu Ser Tyr Pro Asn Asp Asp Leu Glu Ser Leu Asn Tyr
385 390 395 400
Leu His Thr Tyr Ile Ser Phe Ser Tyr Arg Ser Asn Lys Pro Tyr Leu
405 410 415
Ser Val Tyr Leu His Thr Phe Glu Ser Gly His Trp Pro Ile Phe Pro
420 425 430
Asp Ser Pro Thr Ala Phe Asp Ala Tyr Arg Arg Cys Asp Met Ala Ser
435 440 445
Lys Lys Ile Asn Leu Ala Asp Tyr
450 455
<210> 26
<211> 450
<212> PRT
<213> Neotyphodium coenophialum
<400> 26
Met Val Leu Ala Lys Thr Leu His Gln Glu Val Tyr Gln Thr Leu Ser
1 5 10 15
Glu Thr Phe Asp Phe Ala Asn Asn Asp Gln Arg Leu Trp Trp His Ser
20 25 30
Thr Ala Pro Met Phe Gln Lys Ile Leu Gln Thr Ala Asn Tyr Ser Ile
35 40 45
Tyr Ala Gln Tyr Gln His Leu Ser Ile Tyr Lys Ser His Ile Ile Pro
50 55 60
Phe Leu Gly Val Tyr Pro Thr Arg Ser Gly Glu Arg Trp Leu Ser Ile
65 70 75 80
Leu Thr Arg Tyr Gly Thr Pro Phe Glu Leu Ser Leu Asn Cys Ser Asp
85 90 95
Ser Ile Val Arg Tyr Thr Tyr Glu Pro Ile Asn Ala Ala Thr Gly Ser
100 105 110
His Leu Asp Pro Phe Asn Thr Phe Ala Ile Trp Glu Ala Leu Lys Lys
115 120 125
Leu Ile Asp Ser Gln Pro Gly Ile Asp Leu Gln Trp Phe Ser Tyr Phe
130 135 140
Lys Gln Glu Leu Thr Leu Asp Ala Asn Glu Ser Thr Tyr Leu His Ser
145 150 155 160
Gln Asn Leu Val Lys Glu Gln Ile Lys Thr Gln Asn Lys Leu Ala Leu
165 170 175
Asp Leu Lys Gly Asp Lys Phe Val Leu Lys Thr Tyr Ile Tyr Pro Glu
180 185 190
Leu Lys Ser Val Ala Thr Gly Lys Ser Val Gln Glu Leu Val Phe Gly
195 200 205
Ser Val Arg Lys Leu Ala Gln Lys His Lys Ser Ile Arg Pro Ala Phe
210 215 220
Glu Met Leu Glu Asp Tyr Val Gln Ser Arg Asn Lys Val Pro Thr Thr
225 230 235 240
Asp Asp Ser His Asn Thr Pro Leu Ser Ser Arg Leu Leu Ser Cys Asp
245 250 255
Leu Val Ser Pro Thr Lys Ser Arg Val Lys Ile Tyr Leu Leu Glu Arg
260 265 270
Met Val Ser Leu Pro Ala Met Glu Asp Leu Trp Thr Leu Gly Gly Arg
275 280 285
Arg Glu Asp Gln Ser Thr Ile Glu Gly Leu Glu Met Ile Arg Glu Leu
290 295 300
Trp Gly Leu Leu Asn Met Ser Pro Gly Leu Arg Ala Tyr Pro Glu Pro
305 310 315 320
Tyr Leu Pro Leu Gly Ala Ile Pro Asn Glu Gln Leu Pro Ser Met Ala
325 330 335
Asn Tyr Thr Leu His His Asn Asp Pro Ile Pro Glu Pro Gln Val Tyr
340 345 350
Phe Thr Val Phe Gly Met Asn Asp Met Glu Val Thr Asn Ala Leu Thr
355 360 365
Lys Phe Phe Met Arg His Glu Trp Ser Asp Met Ala Ser Lys Tyr Lys
370 375 380
Ala Cys Leu Arg Glu Ser Phe Pro His His Asn Tyr Glu Ala Leu Asn
385 390 395 400
Tyr Ile His Ser Tyr Ile Ser Phe Ser Tyr Arg Asn Asn Lys Pro Tyr
405 410 415
Leu Ser Val Tyr Leu His Ser Phe Glu Thr Gly Glu Trp Pro Val Phe
420 425 430
Pro Glu Gly Leu Ile Ala Phe Asp Gly Cys Arg Arg Asp Leu Thr Cys
435 440 445
Tyr Lys
450
<210> 27
<211> 465
<212> PRT
<213> Balansia obtecta
<400> 27
Met Ala Thr Glu Asn Gly Pro Asp Lys Ser Val Tyr Glu Thr Leu Ser
1 5 10 15
Leu Ile Phe Asn Phe Pro Asp Asp Thr Gln Arg Leu Trp Trp His Ser
20 25 30
Thr Ala Pro Met Leu Ala Glu Met Leu Gln Thr Cys Glu Tyr Ser Val
35 40 45
His Asn Gln Tyr Gln Gln Leu Gly Ile Phe Lys Lys His Ile Ile Pro
50 55 60
Tyr Leu Gly Val Tyr Pro Thr Lys Ser Lys Glu Arg Trp Leu Ser Ile
65 70 75 80
Leu Thr Arg Tyr Gly Ile Pro Phe Glu Leu Ser Leu Asn Cys Ser Asp
85 90 95
Ser Val Val Arg Tyr Thr Tyr Glu Pro Ile Asn Glu Ala Thr Gly Thr
100 105 110
Glu Lys Asp Pro Tyr Asn Thr Leu Ala Ile Leu Asp Ser Ala Gln Glu
115 120 125
Leu Ile Arg Ile Gln Ala Gly Ile Asp Leu Glu Trp Phe Thr Tyr Phe
130 135 140
Lys Asp Glu Leu Thr Val Asn Met Thr Glu Ser Glu Tyr Leu Arg Ser
145 150 155 160
Asn Gly Leu Val Asn Cys Gln Ile Lys Thr Gln Asn Lys Leu Ala Leu
165 170 175
Asp Leu Lys Gly Asp Gln Phe Thr Leu Lys Val Tyr Met Tyr Pro Glu
180 185 190
Leu Lys Ser Val Ala Thr Gly Lys Ser Leu His Glu Leu Ile Phe Gly
195 200 205
Ser Val Arg Arg Leu Ser Leu Lys Tyr Asn Ser Ile Arg Ala Pro Leu
210 215 220
Asp Met Leu Asp Asp Tyr Val Thr Ser Arg Asn Met Gly Ala Asn Gly
225 230 235 240
Glu Glu His Leu Pro Leu Glu Pro Arg Leu Leu Ser Cys Asp Leu Met
245 250 255
Asp Pro His Lys Ser Arg Val Lys Ile Tyr Leu His Glu Arg Lys Val
260 265 270
Ser Leu Ser Ala Met Glu Asp Leu Trp Thr Leu Gly Gly Arg Arg Thr
275 280 285
Asp Ser Thr Thr Met Asp Gly Leu Asn Met Val Arg Glu Leu Trp Asp
290 295 300
Leu Leu Glu Ile Pro Thr Cys Leu Gln Lys Tyr Pro Ala Pro Phe Leu
305 310 315 320
Glu Leu Gly Gln Ile Pro Arg Glu Gln Leu Pro Ser Met Ala Asn Tyr
325 330 335
Thr Leu His His Gly Asp Pro Met Pro Glu Pro Gln Val Tyr Phe Thr
340 345 350
Val Phe Gly Met Asn Asp Ser Lys Val Ile Ser Ala Leu Thr Glu Phe
355 360 365
Phe Lys Arg Arg Val Trp Asn Gly Met Ala Arg Lys Tyr Arg Ala Phe
370 375 380
Leu Gln Asn Ser Tyr Pro Asn Asp Asp His Glu Ser Leu Asn Tyr Leu
385 390 395 400
His Thr Tyr Ile Ser Phe Ser Tyr Arg Lys Asn Lys Pro Tyr Leu Ser
405 410 415
Val Tyr Leu His Thr Phe Glu Ser Gly Ser Trp Pro Ile Phe Pro Asp
420 425 430
Ser Ser Thr Ala Phe Lys Thr Tyr Arg Arg Cys Asp Leu Ser Cys Lys
435 440 445
Asp Asn Gly Leu Asp Asp Lys Leu Leu Ser Pro Val Cys Gln Leu Arg
450 455 460
Val
465
<210> 28
<211> 448
<212> PRT
<213> Claviceps purpurea
<400> 28
Met Ser Thr Ala Lys Asp Pro Gly Asn Gly Val Tyr Glu Ile Leu Ser
1 5 10 15
Leu Ile Phe Asp Phe Pro Ser Asn Glu Gln Arg Leu Trp Trp His Ser
20 25 30
Thr Ala Pro Met Phe Ala Ala Met Leu Asp Asn Ala Gly Tyr Asn Ile
35 40 45
His Asp Gln Tyr Arg His Leu Gly Ile Phe Lys Lys His Ile Ile Pro
50 55 60
Phe Leu Gly Val Tyr Pro Thr Lys Asp Lys Glu Arg Trp Leu Ser Ile
65 70 75 80
Leu Thr Arg Cys Gly Leu Pro Leu Glu Leu Ser Leu Asn Cys Thr Asp
85 90 95
Ser Val Val Arg Tyr Thr Tyr Glu Pro Ile Asn Glu Val Thr Gly Thr
100 105 110
Glu Lys Asp Pro Phe Asn Thr Leu Ala Ile Met Ala Ser Val Gln Lys
115 120 125
Leu Ala Gln Ile Gln Ala Gly Ile Asp Leu Glu Trp Phe Ser Tyr Phe
130 135 140
Lys Asp Glu Leu Thr Leu Asp Glu Ser Glu Ser Ala Thr Leu Gln Ser
145 150 155 160
Asn Glu Leu Val Lys Glu Gln Ile Lys Thr Gln Asn Lys Leu Ala Leu
165 170 175
Asp Leu Lys Glu Ser Gln Phe Ala Leu Lys Val Tyr Phe Tyr Pro His
180 185 190
Leu Lys Ser Ile Ala Thr Gly Lys Ser Thr His Asp Leu Ile Phe Asp
195 200 205
Ser Val Phe Lys Leu Ser Gln Lys His Asp Ser Ile Gln Pro Ala Phe
210 215 220
Gln Val Leu Cys Asp Tyr Val Ser Arg Arg Asn His Ser Ala Glu Ser
225 230 235 240
Asp Gln His Ile Ala Leu His Ala Arg Leu Leu Ser Cys Asp Leu Ile
245 250 255
Asp Pro Ala Lys Ser Arg Val Lys Ile Tyr Leu Leu Glu Lys Thr Val
260 265 270
Ser Leu Ser Val Met Glu Asp Leu Trp Thr Leu Gly Gly Gln Arg Val
275 280 285
Asp Ala Ser Thr Met Asp Gly Leu Asp Met Leu Arg Glu Leu Trp Ser
290 295 300
Leu Leu Lys Val Pro Thr Gly His Leu Glu Tyr Pro Lys Gly Tyr Leu
305 310 315 320
Glu Leu Gly Glu Ile Pro Asn Glu Gln Leu Pro Ser Met Ala Asn Tyr
325 330 335
Thr Leu His His Asn Asn Pro Met Pro Glu Pro Gln Val Tyr Phe Thr
340 345 350
Val Phe Gly Met Asn Asp Ala Glu Ile Ser Asn Ala Leu Thr Ile Phe
355 360 365
Phe Gln Arg His Gly Phe Asp Asp Met Ala Lys Lys Tyr Arg Val Phe
370 375 380
Leu Gln Asp Ser Tyr Pro Tyr His Asp Phe Glu Ser Leu Asn Tyr Leu
385 390 395 400
His Ala Tyr Ile Ser Phe Ser Tyr Arg Arg Asn Lys Pro Tyr Leu Ser
405 410 415
Val Tyr Leu His Thr Phe Glu Thr Gly Arg Trp Pro Val Val Ala Asp
420 425 430
Ser Pro Ile Ser Phe Asp Ala Tyr Arg Arg Cys Asp Leu Ser Thr Lys
435 440 445
<210> 29
<211> 306
<212> PRT
<213> Penicillium roqueforti
<400> 29
Ser Met Ala Pro Met Phe Ala Glu Met Leu Lys Val Ala Gly Tyr Asp
1 5 10 15
Ile His Ser Arg Tyr Lys Ala Leu Gly Leu Tyr Gln Lys Phe Ile Ile
20 25 30
Pro Phe Leu Gly Val Tyr Pro Thr Lys Thr Asn Asp Arg Trp Leu Ser
35 40 45
Ile Leu Thr Arg Tyr Gly Thr Pro Phe Glu Leu Ser Leu Asn Cys Thr
50 55 60
His Ser Val Val Arg Tyr Thr Phe Glu Pro Ile Asn Ala Ala Thr Gly
65 70 75 80
Ser Leu Lys Asp Pro Phe Asn Thr His Ala Ile Trp Asp Ala Leu Asp
85 90 95
Thr Leu Ile Pro Leu Gln Lys Gly Ile Asp Leu Glu Phe Phe Ala His
100 105 110
Leu Lys Arg Asp Leu Thr Val Asn Asp Gln Asp Thr Ala Tyr Leu Leu
115 120 125
Glu Lys Lys Lys Val Gly Gly Gln Ile Arg Thr Gln Asn Lys Leu Ala
130 135 140
Leu Asp Leu Lys Gly Gly Glu Phe Val Leu Lys Ala Tyr Ile Tyr Pro
145 150 155 160
Ala Leu Lys Ser Leu Ala Thr Gly Lys Pro Val Gln Glu Leu Met Phe
165 170 175
Asp Ser Val His Arg Leu Ser His Gln Tyr Pro Thr Leu Ala Ala Pro
180 185 190
Leu Arg Lys Leu Glu Glu Tyr Val His Ser Arg Gly Thr Ser Ser Thr
195 200 205
Ala Ser Pro Arg Leu Ile Ser Cys Asp Leu Cys Asp Pro Arg Gln Ser
210 215 220
Arg Ile Lys Ile Tyr Leu Leu Glu Leu Asn Val Ser Leu Glu Ser Met
225 230 235 240
Glu Asp Leu Trp Thr Leu Gly Gly Arg Arg Asn Asp Thr Gln Thr Leu
245 250 255
Ala Gly Leu Glu Met Ile Arg Glu Leu Trp Asp Leu Ile Asn Leu Pro
260 265 270
Thr Gly Ile Leu Ser Tyr Pro Glu Pro Tyr Leu Lys Leu Gly Glu Val
275 280 285
Pro Asn Glu Gln Leu Pro Leu Met Ala Asn Tyr Thr Leu His His Asp
290 295 300
Asp Pro
305
<210> 30
<211> 329
<212> PRT
<213> Arthroderma gypseum
<400> 30
Met Pro Leu Gln Lys Gly Ile Asp Leu Glu Phe Phe Lys His Leu Lys
1 5 10 15
Gln Asp Leu Thr Val Asp Asp Gln Asp Ser Ala Tyr Leu Leu Glu Asn
20 25 30
Asn Leu Val Gly Gly Gln Ile Arg Thr Gln Asn Lys Leu Ala Leu Asp
35 40 45
Leu Lys Gly Gly Asn Phe Val Leu Lys Thr Tyr Ile Tyr Pro Ala Leu
50 55 60
Lys Ala Leu Ala Thr Gly Lys Ser Ile Lys Thr Leu Met Phe Asp Ser
65 70 75 80
Val Tyr Arg Leu Cys Arg Gln Asn Pro Ser Leu Glu Ala Pro Leu Arg
85 90 95
Val Leu Glu Glu Tyr Val Asp Ser Lys Gly Pro Asn Ser Thr Ala Ser
100 105 110
Pro Arg Leu Leu Ser Cys Asp Leu Ile Asp Pro Ser Lys Ser Arg Val
115 120 125
Lys Ile Tyr Ile Leu Glu Leu Asn Val Thr Leu Glu Ala Met Glu Asp
130 135 140
Leu Trp Thr Leu Gly Gly Arg Leu Asn Asp Ala Ala Thr Leu Ala Gly
145 150 155 160
Leu Glu Met Val Arg Glu Leu Trp Asp Leu Val Lys Leu Pro Pro Gly
165 170 175
Leu Arg Asp Tyr Pro Glu Pro Phe Leu Gln Leu Gly Thr Ile Pro Asp
180 185 190
Glu Gln Leu Pro Leu Met Ala Asn Tyr Thr Leu His His Asn Glu Ala
195 200 205
Met Pro Glu Pro Gln Val Tyr Phe Thr Thr Phe Gly Leu Asn Asp Gly
210 215 220
Arg Ile Ala Glu Gly Leu Val Thr Phe Phe Glu Arg Arg Gly Trp Asp
225 230 235 240
His Met Ala Arg Thr Tyr Lys Asp Ser Leu Arg Ala Tyr Tyr Pro His
245 250 255
Ala Asp Gln Glu Thr Leu Asn Tyr Leu His Ala Tyr Ile Ser Phe Ser
260 265 270
Tyr Arg Lys Gly Thr Pro Tyr Leu Ser Val Tyr Leu Gln Thr Phe Glu
275 280 285
Thr Gly Asp Trp Pro Ile Ser Asn Phe Gly Ile Pro Val Val Lys Pro
290 295 300
Leu Arg Ser Asn Val Gly Cys Gln His Pro Ile Ser Phe Ser Ile Pro
305 310 315 320
Ile Thr Lys Ala Pro Leu Leu Gly Val
325
<210> 31
<211> 379
<212> PRT
<213> Paecilomyces divaricatus
<400> 31
Met Thr Gln Ala Ser Pro Ala Ser Lys Leu Phe Thr Pro Leu Ser Val
1 5 10 15
Gly Ser Cys Lys Leu Tyr His Arg Leu Val Met Ala Pro Met Thr Arg
20 25 30
Phe Arg Ala Asp Glu Ser Ala Ile Gln Leu Pro Phe Val Lys Glu Tyr
35 40 45
Tyr Gly Gln Arg Ala Ser Val Pro Gly Thr Leu Leu Ile Ser Glu Ala
50 55 60
Thr Asp Ile Ser Pro Gln Ala Gly Gly Phe Ala Asn Val Pro Gly Ile
65 70 75 80
Trp Asn Glu Ala Gln Ile Gln Ala Trp Arg Gln Ile Val Gly Glu Val
85 90 95
His Ser Lys Gly Ser Phe Ile Phe Cys Gln Leu Trp Ala Thr Gly Arg
100 105 110
Ala Ala Glu Pro Ser Val Leu Ala Glu Lys Gly Phe Thr Leu Val Ser
115 120 125
Ser Ser Ala Thr Pro Ile Ser Pro Asp Gly Leu Leu Pro Cys Ala Leu
130 135 140
Thr Glu Val Glu Ile His Asp Tyr Ile Asp Asp Phe Ala Gln Ala Ala
145 150 155 160
Gln Asn Ala Ile Leu Ala Gly Phe Asp Gly Val Glu Ile His Gly Ala
165 170 175
Asn Gly Tyr Leu Ile Asp Gln Phe Thr Gln Ala Ser Cys Asn Lys Arg
180 185 190
Ile Asp His Trp Gly Gly Ser Ile Ala Asn Arg Ala Arg Phe Ala Ile
195 200 205
Glu Val Thr Arg Ala Val Val Ala Ala Val Gly Ala Asp Arg Val Gly
210 215 220
Val Lys Leu Ser Pro Trp Ser Gln Tyr Gln Gly Met Gly Asn Met Asp
225 230 235 240
Gly Leu Val Ala Gln Phe Glu Tyr Leu Ile Ser Gln Leu Gln Lys Met
245 250 255
Asp Leu Ala Tyr Leu His Leu Ala Asn Ser Thr Trp Leu Gly Glu Glu
260 265 270
Gly Pro His Ser Asp Pro His His Glu Val Phe Val Gln Ala Trp Gly
275 280 285
Lys Ser Lys Pro Ile Leu Leu Ala Gly Gly Tyr Asp Ala Ala Ser Ala
290 295 300
Lys Gln Val Val Asp Met Leu Tyr Gly Glu His Asp Asn Val Ala Ile
305 310 315 320
Ala Phe Gly Arg Phe Phe Ile Ser Asn Pro Asp Leu Pro Phe Arg Val
325 330 335
Lys Ser Asn Val Lys Leu Gln Asp Tyr Ser Arg Ser Ser Met Asp Thr
340 345 350
Ser Val Ser Lys Asp Gly Tyr Leu Asp His Pro Phe Ser Pro Glu Phe
355 360 365
Leu Ala Ser Asn Arg Ser Glu Phe Ala Ser Thr
370 375
<210> 32
<211> 376
<212> PRT
<213> Aspergillus fumigatus
<400> 32
Met Arg Glu Glu Pro Ser Ser Ala Gln Leu Phe Lys Pro Leu Lys Val
1 5 10 15
Gly Arg Cys His Leu Gln His Arg Met Ile Met Ala Pro Thr Thr Arg
20 25 30
Phe Arg Ala Asp Gly Gln Gly Val Pro Leu Pro Phe Val Gln Glu Tyr
35 40 45
Tyr Gly Gln Arg Ala Ser Val Pro Gly Thr Leu Leu Ile Thr Glu Ala
50 55 60
Thr Asp Ile Thr Pro Lys Ala Met Gly Tyr Lys His Val Pro Gly Ile
65 70 75 80
Trp Ser Glu Pro Gln Arg Glu Ala Trp Arg Glu Ile Val Ser Arg Val
85 90 95
His Ser Lys Lys Cys Phe Ile Phe Cys Gln Leu Trp Ala Thr Gly Arg
100 105 110
Ala Ala Asp Pro Asp Val Leu Ala Asp Met Lys Asp Leu Ile Ser Ser
115 120 125
Ser Ala Val Pro Val Glu Glu Lys Gly Pro Leu Pro Arg Ala Leu Thr
130 135 140
Glu Asp Glu Ile Gln Gln Cys Ile Ala Asp Phe Ala Gln Ala Ala Arg
145 150 155 160
Asn Ala Ile Asn Ala Gly Phe Asp Gly Val Glu Ile His Gly Ala Asn
165 170 175
Gly Tyr Leu Ile Asp Gln Phe Thr Gln Lys Ser Cys Asn His Arg Gln
180 185 190
Asp Arg Trp Gly Gly Ser Ile Glu Asn Arg Ala Arg Phe Ala Val Glu
195 200 205
Leu Thr Arg Ala Val Ile Glu Ala Val Gly Ala Asp Arg Val Gly Val
210 215 220
Lys Leu Ser Pro Tyr Ser Gln Tyr Leu Gly Met Gly Thr Met Asp Glu
225 230 235 240
Leu Val Pro Gln Phe Glu Tyr Leu Ile Ala Gln Met Arg Arg Leu Asp
245 250 255
Val Ala Tyr Leu His Leu Ala Asn Ser Arg Trp Leu Asp Glu Glu Lys
260 265 270
Pro His Pro Asp Pro Asn His Glu Val Phe Val Arg Val Trp Gly Gln
275 280 285
Ser Ser Pro Ile Leu Leu Ala Gly Gly Tyr Asp Ala Ala Ser Ala Glu
290 295 300
Lys Val Thr Glu Gln Met Ala Ala Ala Thr Tyr Thr Asn Val Ala Ile
305 310 315 320
Ala Phe Gly Arg Tyr Phe Ile Ser Thr Pro Asp Leu Pro Phe Arg Val
325 330 335
Met Ala Gly Ile Gln Leu Gln Lys Tyr Asp Arg Ala Ser Phe Tyr Ser
340 345 350
Thr Leu Ser Arg Glu Gly Tyr Leu Asp Tyr Pro Phe Ser Ala Glu Tyr
355 360 365
Met Ala Leu His Asn Phe Pro Val
370 375
<210> 33
<211> 380
<212> PRT
<213> Neotyphodium lolii
<400> 33
Met Ser Thr Ser Asn Leu Phe Thr Pro Leu Gln Phe Gly Lys Cys Leu
1 5 10 15
Leu Gln His Lys Leu Val Leu Ser Pro Met Thr Arg Phe Arg Ala Asp
20 25 30
Asn Glu Gly Val Pro Leu Pro Tyr Val Lys Thr Tyr Tyr Cys Gln Arg
35 40 45
Ala Ser Leu Pro Gly Thr Leu Leu Leu Thr Glu Ala Thr Ala Ile Ser
50 55 60
Arg Arg Ala Arg Gly Phe Pro Asn Val Pro Gly Ile Trp Ser Gln Glu
65 70 75 80
Gln Ile Ala Gly Trp Lys Glu Val Val Asp Ala Val His Ala Lys Gly
85 90 95
Ser Tyr Ile Trp Leu Gln Leu Trp Ala Thr Gly Arg Ala Ala Glu Val
100 105 110
Gly Val Leu Lys Ala Asn Gly Phe Asp Leu Val Ser Ser Ser Ala Val
115 120 125
Pro Val Ser Pro Gly Glu Pro Thr Pro Arg Ala Leu Ser Asp Asp Glu
130 135 140
Ile Asn Ser Tyr Ile Gly Asp Phe Val Gln Ala Ala Lys Asn Ala Val
145 150 155 160
Leu Glu Ala Gly Phe Asp Gly Val Glu Leu His Gly Ala Asn Gly Phe
165 170 175
Leu Ile Asp Gln Phe Leu Gln Ser Pro Cys Asn Gln Arg Thr Asp Gln
180 185 190
Trp Gly Gly Cys Ile Glu Asn Arg Ser Arg Phe Gly Leu Glu Ile Thr
195 200 205
Arg Arg Val Ile Asp Ala Val Gly Lys Asp His Val Gly Met Lys Leu
210 215 220
Ser Thr Trp Ser Thr Phe Gln Gly Met Gly Thr Met Asp Asp Leu Ile
225 230 235 240
Pro Gln Phe Glu His Phe Ile Met Arg Leu Arg Glu Ile Gly Ile Ala
245 250 255
Tyr Leu His Leu Ala Asn Ser Arg Trp Val Glu Glu Glu Asp Pro Thr
260 265 270
Ile Arg Thr His Pro Asp Ile His Asn Glu Thr Phe Val Arg Met Trp
275 280 285
Gly Lys Glu Lys Pro Val Leu Leu Ala Gly Gly Tyr Gly Pro Glu Ser
290 295 300
Ala Lys Leu Val Val Asp Glu Thr Tyr Ser Asp His Lys Asn Ile Gly
305 310 315 320
Val Val Phe Gly Arg His Tyr Ile Ser Asn Pro Asp Leu Pro Phe Arg
325 330 335
Leu Lys Met Gly Leu Pro Leu Gln Lys Tyr Asn Arg Glu Thr Phe Tyr
340 345 350
Ile Pro Phe Ser Asp Glu Gly Tyr Leu Asp Tyr Pro Tyr Ser Glu Glu
355 360 365
Tyr Ile Thr Glu Asn Lys Lys Gln Ala Val Leu Ala
370 375 380
<210> 34
<211> 380
<212> PRT
<213> Periglandula ipomoeae
<400> 34
Met Ser Thr Ser Asn Leu Phe Asn Glu Leu Pro Phe Gly Thr Ser Val
1 5 10 15
Leu Lys His Lys Ile Val Leu Ser Pro Met Thr Arg Phe Arg Ala Asp
20 25 30
Asn Gly Gly Val Pro Leu Pro Tyr Val Lys Thr Tyr Tyr Ala Gln Arg
35 40 45
Ala Ser Val His Gly Thr Leu Leu Ile Thr Glu Ala Val Ala Ile Cys
50 55 60
Pro Arg Ala Lys Gly Phe Pro Asn Ile Pro Gly Ile Trp His Asp Ser
65 70 75 80
Gln Ile Ala Ala Trp Lys Gly Val Val Asp Glu Val His Ser Lys Gly
85 90 95
Ser Val Ile Trp Leu Gln Leu Trp Ala Thr Gly Arg Ala Val Asp Pro
100 105 110
Asp Thr Leu Thr Ser Asn Gly Leu Glu Leu Lys Ser Ser Ser Glu Val
115 120 125
Pro Val Ala Pro Gly Glu Pro Thr Pro Arg Pro Leu Ser Glu Glu Glu
130 135 140
Ile Gln Ser Tyr Ile Phe Asp Tyr Ala Gln Gly Ala Lys Asn Ala Val
145 150 155 160
His Glu Ala Gly Phe Asp Gly Val Glu Ile His Gly Ala Asn Gly Phe
165 170 175
Leu Ile Asp Gln Phe Leu Gln Ser Ser Cys Asn Arg Arg Thr Asp Gln
180 185 190
Trp Gly Gly Ser Ile Glu Asn Arg Ser Arg Phe Gly Leu Glu Ile Thr
195 200 205
Arg Arg Val Ile Asp Ala Val Gly Asn Asp His Val Gly Val Lys Leu
210 215 220
Ser Pro Trp Ser Thr Phe Gln Gly Met Gly Thr Met Asp Asp Leu Val
225 230 235 240
Pro Gln Phe Glu His Phe Ile Thr Arg Leu Arg Glu Met Asp Ile Ala
245 250 255
Tyr Leu His Leu Ala Asn Ser Arg Trp Val Glu Glu Glu Asp Pro Ser
260 265 270
Ile Lys Thr His Pro Asp Val His Asn Glu Thr Phe Val Arg Ile Trp
275 280 285
Gly Glu Lys Arg Pro Ile Leu Leu Ala Gly Gly Tyr Asn Pro Asp Ser
290 295 300
Ala Arg Arg Leu Val Asp Gln Thr Tyr Ser Glu Gln Asn Asn Val Met
305 310 315 320
Val Val Phe Gly Arg His Tyr Ile Ser Asn Pro Asp Leu Pro Phe Arg
325 330 335
Leu Lys Met Gly Ile Ala Leu Gln Arg Tyr Asn Arg Asp Thr Phe Tyr
340 345 350
Ile Pro Phe Ser Gly Glu Gly Tyr Leu Asp Tyr Pro Phe Cys Lys Glu
355 360 365
Tyr Leu Lys Gln Gly Asp Gln Glu Thr Thr Val Ala
370 375 380
<210> 35
<211> 382
<212> PRT
<213> Claviceps fusiformis
<400> 35
Met Ser Ser Ser Asn Leu Phe Lys Pro Ile Pro Leu Gly Arg Lys Val
1 5 10 15
Leu Gln His Lys Val Val Leu Ser Pro Met Thr Arg Phe Arg Ala Asp
20 25 30
Asn Asp Gly Val Pro Leu Ser Tyr Val Lys Ser Tyr Tyr Gly Gln Arg
35 40 45
Ala Ser Ile Arg Gly Thr Leu Leu Ile Thr Glu Ala Val Ala Ile Cys
50 55 60
Pro Arg Ala Lys Gly Phe Ser Asn Cys Pro Gly Ile Trp His Gln Asp
65 70 75 80
Gln Ile Ala Ala Trp Lys Glu Val Val Asp Glu Val His Ser Lys Gly
85 90 95
Ser Val Ile Trp Leu Gln Leu Trp Ala Thr Gly Arg Ala Ser Asp Ala
100 105 110
Asp Thr Leu Lys Glu Ser Gly Phe His Leu Glu Ser Ser Ser Asp Val
115 120 125
Pro Val Ala Pro Gly Glu Pro Val Pro Arg Pro Leu Ser Glu Asp Glu
130 135 140
Ile Glu Ser Tyr Ile Arg Asp Tyr Val Thr Gly Ala Ile Asn Ala Val
145 150 155 160
Gln Gly Ala Gly Phe Asp Gly Ile Glu Ile His Gly Ala Asn Gly Phe
165 170 175
Leu Val Asp Gln Phe Leu Gln Ala Ser Cys Asn Thr Arg Ala Asp Gln
180 185 190
Trp Gly Gly Ser Ile Glu Asn Arg Ser Arg Phe Gly Leu Glu Ile Thr
195 200 205
Arg Arg Val Val Asp Ala Val Gly Lys Asp Arg Val Gly Val Lys Leu
210 215 220
Ser Pro Trp Ser Thr Phe Gln Gly Met Gly Thr Met Asp Asp Leu Val
225 230 235 240
Ala Gln Phe Glu His Phe Ile Ser Arg Leu Arg Glu Met Asp Ile Ala
245 250 255
Tyr Ile His Leu Val Asn Thr Arg Trp Leu Glu Glu Glu Glu Pro Gly
260 265 270
Ile Lys Thr His Pro Asp Val Asp Asn Gln Thr Phe Val Arg Met Trp
275 280 285
Gly Asn Lys Thr Pro Ile Leu Leu Ala Gly Gly Tyr Asp Ala Asp Ser
290 295 300
Ala Arg Arg Leu Val Asp Glu Thr Tyr Ser Asp Gln Asn Asn Ile Met
305 310 315 320
Val Val Phe Gly Arg His Tyr Ile Ser Asn Pro Asp Leu Pro Phe Arg
325 330 335
Leu Arg Leu Gly Ile Pro Leu Gln Lys Tyr Asn Arg Asp Thr Phe Tyr
340 345 350
Ile Pro Phe Ser Asp Glu Gly Tyr Leu Asp Tyr Pro Phe Cys Gln Glu
355 360 365
Phe Leu Asp Gln Gln Asp Val Asp Gln Val Val Val Ala Ala
370 375 380
<210> 36
<211> 368
<212> PRT
<213> Penicillium chrysogenum
<400> 36
Met Thr Lys Leu Phe Thr Pro Leu Arg Val Gly Arg Ala Glu Leu Ser
1 5 10 15
Asn Arg Ile Ala Met Ala Pro Met Thr Arg Phe Arg Ala Asp Asp Asn
20 25 30
His Thr Pro Leu Pro Ile Val Lys Asp Tyr Tyr Ala Gln Arg Ala Ser
35 40 45
Val Pro Gly Thr Leu Leu Ile Thr Glu Ala Thr Phe Ile Ser Ala Arg
50 55 60
Ala Gly Gly Tyr Pro Asn Val Pro Gly Ile Tyr Asn Asp Ala Gln Ile
65 70 75 80
Ala Ala Trp Lys Glu Val Thr Asp Ala Val His Ala Lys Gly Ser Tyr
85 90 95
Ile Tyr Leu Gln Leu Trp Ala Leu Gly Arg Val Ala Ser Val Glu Leu
100 105 110
Leu Gln Ala Ser Gly Phe Asp Leu Val Ser Ser Ser Ala Thr Pro Ala
115 120 125
Ser Ala Asp Ala Pro Val Pro Arg Ala Leu Thr Glu Ser Glu Ile His
130 135 140
Glu Trp Ile Ala Asp Tyr Ala Gln Ala Ala Arg Asn Ala Val Ala Ala
145 150 155 160
Gly Phe Asp Gly Val Glu Ile His Ala Ala Asn Gly Tyr Leu Ile Asp
165 170 175
Gln Phe Thr Gln Asp Thr Cys Asn Thr Arg Asp Asp Ala Trp Gly Gly
180 185 190
Ser Val Glu Gly Arg Ala Arg Phe Ala Val Glu Val Ser Arg Ala Val
195 200 205
Val Glu Ala Val Gly Ala Asp Arg Thr Gly Ile Arg Phe Ser Pro Trp
210 215 220
Ser Thr Phe Gln Gly Met Arg Met Glu Glu Pro Lys Pro Gln Phe Glu
225 230 235 240
Tyr Leu Ala Glu Arg Met Ala Glu Met Gly Leu Ala Tyr Val His Leu
245 250 255
Val Glu Ser Arg Ile Ala Gly Asn Ala Glu Val Glu Ala Asp Asp Asp
260 265 270
Leu Asp Phe Phe Leu Lys Ala Tyr Gly Lys Ala Ser Pro Val Ile Val
275 280 285
Ala Gly Gly Tyr Val Ala Asp Ser Ala Leu Gln Ala Ala Asp Val Lys
290 295 300
Tyr Ala Glu Tyr Asp Ala Ile Val Gly Ile Gly Arg Pro Phe Ile Ser
305 310 315 320
Asn Pro Asp Leu Pro Phe Arg Val Gln Lys Gly Ile Pro Phe Val Pro
325 330 335
Tyr Asp Arg Asp Thr Phe Tyr Val Pro Lys Asp Pro Lys Gly Tyr Ile
340 345 350
Asp Tyr Ala Phe Ser Ala Glu Phe Gln Lys Ser Ile Glu Ala Ala Ala
355 360 365
<210> 37
<211> 369
<212> PRT
<213> Claviceps purpurea
<400> 37
Met Ser Thr Ser Asn Leu Phe Ser Thr Val Pro Phe Gly Lys Asn Val
1 5 10 15
Leu Asn His Lys Ile Val Leu Ser Pro Met Thr Arg Phe Arg Ala Asp
20 25 30
Asp Asn Gly Val Pro Leu Ser Tyr Met Lys Thr Phe Tyr Ala Gln Arg
35 40 45
Ala Ser Val Arg Gly Thr Leu Leu Val Thr Asp Ala Val Ala Ile Cys
50 55 60
Pro Arg Thr Lys Gly Phe Pro Asn Val Pro Gly Ile Trp His Lys Asp
65 70 75 80
Gln Ile Ala Ala Trp Lys Glu Val Val Asp Glu Val His Ser Lys Gly
85 90 95
Ser Phe Ile Trp Leu Gln Leu Trp Ala Thr Gly Arg Ala Ala Asp Leu
100 105 110
Glu Ala Leu Thr Ser Gln Gly Leu Lys Leu Glu Ser Ser Ser Glu Val
115 120 125
Pro Val Ala Pro Gly Glu Pro Thr Pro Arg Ala Leu Asp Glu Asp Glu
130 135 140
Ile Gln Gln Tyr Ile Leu Asp Tyr Val Gln Gly Ala Lys Asn Ala Val
145 150 155 160
His Gly Ala Gly Phe Asp Gly Val Glu Ile His Gly Ala Asn Gly Phe
165 170 175
Leu Ile Asp Gln Phe Leu Gln Ser Ser Cys Asn Arg Arg Thr Asp Gln
180 185 190
Trp Gly Gly Ser Ile Glu Asn Arg Ser Arg Phe Gly Leu Glu Ile Thr
195 200 205
Arg Gly Val Val Asp Ala Val Gly His Asp Arg Val Gly Met Lys Leu
210 215 220
Ser Pro Trp Ser Thr Phe Gln Gly Met Gly Thr Met Asp Asp Leu Val
225 230 235 240
Pro Gln Phe Glu His Phe Ile Thr Cys Leu Arg Glu Met Asp Ile Ala
245 250 255
Tyr Leu His Leu Ala Asn Ser Arg Trp Val Glu Glu Glu Asp Pro Ser
260 265 270
Ile Arg Thr His Pro Asp Phe His Asn Gln Thr Phe Val Gln Met Trp
275 280 285
Gly Lys Lys Arg Pro Ile Leu Leu Ala Gly Gly Tyr Asp Pro Asp Ser
290 295 300
Ala Arg Arg Leu Val Asp Gln Thr Tyr Ser Asp Arg Asn Asn Val Leu
305 310 315 320
Val Val Phe Gly Arg His Tyr Ile Ser Asn Pro Asp Leu Pro Phe Arg
325 330 335
Leu Arg Met Gly Ile Ala Cys Arg Ser Thr Ile Glu Thr His Ser Ile
340 345 350
Phe Pro Ala Arg Glu Arg Ala Met Trp Thr Ile Pro Ser Val Lys Asn
355 360 365
Ile
<210> 38
<211> 370
<212> PRT
<213> Aspergillus flavus
<400> 38
Met Ala Ser Lys Leu Phe Ser Pro Leu Gln Val Gly Arg Met Gln Leu
1 5 10 15
Ala His Arg Ile Thr Met Ala Pro Leu Thr His Phe Arg Asn Asp Asp
20 25 30
Asp His Val Pro Leu Pro Ile Val Lys Glu His Tyr Glu Gln Arg Gly
35 40 45
Ser Val Pro Gly Thr Leu Leu Ile Thr Glu Ala Thr Leu Ile Ser Pro
50 55 60
Arg Ala Gly Gly Tyr Pro Asn Val Pro Gly Ile Trp Ser Glu Ala Gln
65 70 75 80
Ile Ala Ala Trp Arg Thr Val Thr Asp Ala Val His Ala Lys Gly Ser
85 90 95
Tyr Ile Phe Met Gln Leu Trp Ala Leu Gly Arg Val Ala Asn Pro Ala
100 105 110
Asn Leu Gln Lys Ala Gly Tyr Asp Leu Val Ser Ser Ser Ala Val Pro
115 120 125
Ala Thr Pro Glu Gly Ala Val Pro Arg Ala Leu Thr Glu Asp Glu Ile
130 135 140
Arg Asp Tyr Ile Arg Asp Tyr Ala Gln Ala Ala Lys Asn Ala Ile Ala
145 150 155 160
Ala Gly Phe Asp Gly Val Glu Ile His Gly Ala Asn Gly Tyr Leu Ile
165 170 175
Asp Gln Phe Thr Gln Asp Thr Val Asn Gln Arg Thr Asp Ser Trp Gly
180 185 190
Gly Ser Val Glu Asn Arg Ala Arg Phe Ala Leu Glu Val Thr Lys Ala
195 200 205
Val Thr Glu Ala Ile Gly Ala Glu Arg Thr Gly Ile Arg Phe Ser Pro
210 215 220
Phe Ser Thr Phe Gln Gly Met Arg Met Ala Asp Pro Val Pro Gln Phe
225 230 235 240
Glu Tyr Leu Ala Gln Lys Thr Lys Glu Phe Lys Leu Ala Tyr Val His
245 250 255
Leu Val Glu Pro Arg Ile Ala Gly Asn Thr Glu Val Asp Glu Pro Ser
260 265 270
Ala Asp Ser Leu Asp Phe Phe Phe Arg Ala Tyr Glu Lys Ala Gly Pro
275 280 285
Ile Met Val Ala Gly Gly Tyr Lys Ala Glu Ser Ala Lys Glu Ala Val
290 295 300
Asp Ser Gln Tyr Lys Asp Tyr Asp Thr Leu Val Ala Met Gly Arg Pro
305 310 315 320
Phe Thr Ser Asn Pro Asp Leu Pro Phe Lys Val Lys Ala Gly Ile Pro
325 330 335
Leu Arg Ala Tyr Glu Arg Glu Lys Phe Tyr Leu Val Lys Asp Pro Lys
340 345 350
Gly Tyr Thr Asp Tyr Glu Phe Ser Glu Glu Phe Lys Ser Ala Gln Val
355 360 365
Ala Ala
370
<210> 39
<211> 380
<212> PRT
<213> Neurospora tetrasperma
<400> 39
Met Ala Ala Ala Ala Glu Ser Arg Leu Phe Gln Pro Leu Lys Leu Thr
1 5 10 15
Pro Arg Ile Thr Leu Ala His Arg Leu Ala Met Ala Pro Leu Thr Arg
20 25 30
Phe Arg Ser Asp Asp Glu His Val Pro Ile Val Pro Leu Met Thr Thr
35 40 45
Tyr Tyr Ser Gln Arg Ala Ser Val Pro Gly Thr Leu Leu Val Thr Glu
50 55 60
Ala Thr Phe Ile Ser Pro Ala Ala Gly Gly Tyr Asp Asn Val Pro Gly
65 70 75 80
Ile Tyr Asn Ala Ala Gln Ile Ala Ala Trp Lys Lys Ile Thr Asp Ala
85 90 95
Val His Ala Lys Gly Ser Phe Ile Phe Cys Gln Leu Trp Ser Leu Gly
100 105 110
Arg Ala Ala Asn Pro Glu Val Leu Ala Lys Glu Gly Gly Phe Lys Leu
115 120 125
Lys Ser Ser Ser Ala Val Pro Met Glu Glu Gly Ala Pro Val Pro Glu
130 135 140
Glu Met Thr Val Ala Glu Ile Lys Glu Arg Val Ala Glu Tyr Ala Gly
145 150 155 160
Ala Ala Lys Asn Ala Ile Glu Ala Gly Phe Asp Gly Val Glu Ile His
165 170 175
Gly Ala Asn Gly Tyr Leu Ile Asp Gln Phe Leu Gln Asp Thr Cys Asn
180 185 190
Gln Arg Thr Asp Glu Tyr Gly Gly Ser Val Glu Asn Arg Ser Arg Phe
195 200 205
Ala Leu Glu Val Val Lys Ala Val Val Glu Ala Val Gly Ala Glu Arg
210 215 220
Thr Gly Ile Arg Leu Ser Pro Tyr Ser Thr Phe Gln Gly Met Lys Met
225 230 235 240
Lys Lys Asp Leu Val Pro Gln Phe Glu Asp Val Ile Arg Lys Ile Ser
245 250 255
Gly Phe Gly Leu Ala Tyr Leu His Leu Thr Gln Ser Arg Val Ala Gly
260 265 270
Asn Met Asp Val Gln Pro Gly Glu Asp Glu Glu Ser Leu Ala Phe Ala
275 280 285
Ala Arg Leu Trp Asp Gly Pro Leu Leu Ile Ala Gly Gly Leu Thr Ala
290 295 300
Glu Thr Ala Lys His Leu Val Asp His Glu Phe Pro Glu Lys Asp Val
305 310 315 320
Val Ala Thr Phe Gly Arg His Phe Ile Ser Thr Pro Asp Leu Pro Phe
325 330 335
Arg Ile Lys Glu Gly Ile Glu Leu Asn Pro Tyr Asp Arg Asp Thr Phe
340 345 350
Tyr Leu Pro Lys Ser Pro Val Gly Tyr Ile Asp Gln Pro Phe Ser Lys
355 360 365
Glu Phe Glu Lys Val Tyr Gly Ala Gln Ala Leu Asn
370 375 380
<210> 40
<211> 367
<212> PRT
<213> Aspergillus terreus
<400> 40
Met Ser Ser Ser Lys Leu Phe Glu Pro Leu Arg Val Gly Gln Val Glu
1 5 10 15
Leu Gln His Arg Val Val Met Ala Pro Leu Thr Arg Phe Arg Asn Thr
20 25 30
Glu Asp Asn Leu Pro Leu Pro Ile Val Thr Lys Tyr Tyr Glu Gln Arg
35 40 45
Ala Ser Val Pro Gly Thr Leu Leu Ile Ala Glu Ala Asn Gln Ile Ser
50 55 60
Pro Ala Ala Ala Gly Met Pro His Gly Pro Ala Ile Trp Asn Glu Ala
65 70 75 80
His Val Gln Ala Trp Lys Lys Val Thr Asp Ala Val His Ala Lys Gly
85 90 95
Ser His Ile Tyr Cys Gln Leu Ile Ala Leu Gly Arg Ala Ala His Gly
100 105 110
Pro Thr Leu Gln Lys Tyr Gly Asp Tyr Glu Val Ser Ala Pro Ser Pro
115 120 125
Ile Pro Met Glu Lys Asp Gly Val Pro Pro Lys Glu Leu Thr Glu Ala
130 135 140
Gln Ile Gln Gly Phe Ile Ala Asp Phe Ala Gln Ala Gly Lys Asn Ala
145 150 155 160
Ile Ala Ala Gly Phe Asp Gly Val Glu Val His Gly Ala Asn Gly Tyr
165 170 175
Leu Val Asp Gln Phe Thr Gln Asp Val Thr Asn Gln Arg Thr Asp Gly
180 185 190
Trp Gly Gly Ser Val Ala Asn Arg Ala Arg Phe Ala Ile Glu Val Ala
195 200 205
Arg Ala Leu Val Asp Ala Val Gly Ala Asp Arg Val Ala Phe Arg Leu
210 215 220
Ser Pro Trp Asn Thr Trp Gln Gly Met Lys Met Ala Asp Pro Val Pro
225 230 235 240
Gln Phe Ser Tyr Leu Val Glu Lys Leu Lys Glu Leu Lys Leu Ala Tyr
245 250 255
Leu His Leu Val Glu Ser Arg Val Ile Asn Asn Ile Asp Cys Glu Lys
260 265 270
Gly Glu Gly Leu Glu Phe Leu Leu Asp Ile Trp Gly Lys Thr Ser Pro
275 280 285
Val Leu Val Ala Gly Gly Tyr Thr Pro Glu Asn Cys Arg Glu Val Asp
290 295 300
Glu Gln Tyr Lys Asn Asn Asp Val Ala Val Val Phe Gly Arg His Phe
305 310 315 320
Ile Ser Asn Pro Asp Leu Pro Phe Arg Ile Lys Asn Ser Ile Ala Leu
325 330 335
Gln Lys Tyr Asp Arg Asp Thr Phe Tyr Thr Pro Ile Gln Glu Glu Gly
340 345 350
Tyr Leu Asp Tyr Pro Phe Ser Pro Glu Phe Thr Ala Ala Gln Ala
355 360 365
<210> 41
<211> 519
<212> PRT
<213> Paecilomyces divaricatus
<400> 41
Met Arg Thr Lys Asp Asp Asn Ala Pro Lys Pro Asp Pro Asp Gly Val
1 5 10 15
Pro Ala Tyr Thr Thr Ser Asn Gly Cys Pro Val Met Arg Pro Glu Gly
20 25 30
Leu Pro Ala Asp Gly Arg Lys Leu Leu Leu Asn Asp Ser His Leu Ile
35 40 45
Asp Met Leu Ala His Phe Asn Arg Glu Lys Ile Pro Glu Arg Ala Val
50 55 60
His Ala Lys Gly Ala Gly Ala Tyr Gly Glu Phe Glu Val Thr Glu Asp
65 70 75 80
Ile Ser Asp Ile Cys Asn Ile Asp Met Leu Leu Gly Val Gly Lys Lys
85 90 95
Thr Pro Cys Val Thr Arg Phe Ser Thr Thr Gly Leu Glu Arg Gly Ser
100 105 110
Ser Asp Gly Val Arg Asp Leu Lys Gly Leu Ala Thr Lys Phe Tyr Thr
115 120 125
Lys Gln Gly Asn Trp Asp Trp Val Cys Leu Asn Phe Pro Phe Phe Phe
130 135 140
Ile Arg Asp Pro Met Lys Phe Pro Ser Leu Met His Ala Gln Arg Arg
145 150 155 160
Asp Pro Gln Thr Asn Leu Leu Asn Pro Asn Met Tyr Trp Asp Trp Val
165 170 175
Thr Ser Asn His Glu Ser Leu His Met Val Leu Leu Gln Phe Ser Asp
180 185 190
Phe Gly Thr Met Phe Asn Trp Arg Ser Met Ser Gly Tyr Ile Gly His
195 200 205
Ala Tyr Lys Trp Val Met Pro Asp Gly Ser Tyr Lys Tyr Val His Ile
210 215 220
Phe Leu Ser Ser Asn Gln Gly Pro Asn Phe Thr Asp Gly Gly Pro Asp
225 230 235 240
Lys Lys Asp Asn Gly Leu Glu Pro Asp His Ala Thr Arg Asp Leu Tyr
245 250 255
Glu Ala Ile Glu Ser Gly Asp Tyr Pro Ser Trp Thr Ala Asn Val Gln
260 265 270
Val Val Asp Pro Lys Asp Ala Pro Lys Leu Gly Phe Asn Ile Leu Asp
275 280 285
Ile Thr Lys His Trp Asn Leu Gly Thr Tyr Pro Lys Asp Leu Glu Ala
290 295 300
Ile Pro Ser Arg Pro Phe Gly Lys Leu Thr Leu Asn Arg Asn Pro Gln
305 310 315 320
Asn Tyr Phe Ala Glu Ile Glu Gln Leu Ala Phe Ser Pro Ser Asn Met
325 330 335
Val Pro Gly Val Glu Pro Ser Glu Asp Pro Ile Leu Gln Ala Arg Met
340 345 350
Phe Ala Tyr Pro Asp Ala Gln Arg Tyr Arg Leu Gly Val Asn Asn Gln
355 360 365
His Leu Pro Val Asn Arg Pro Gln His Val Phe Asn Pro Leu Leu Arg
370 375 380
Asp Gly Ala Gly Met Ala Ser Gly Asn Tyr Gly Ser Leu Pro Gly Tyr
385 390 395 400
Val Ser Asp Tyr His Pro Met Lys Phe Gln Lys Pro Thr Pro Gln Asn
405 410 415
Pro Glu Phe Asp Ser Trp Leu Ala Gln Ile Ser Ser Gln Ser Trp Gly
420 425 430
Gln Ser Asn Glu Gln Asp Tyr Lys Phe Ala Arg Glu Tyr Tyr Leu Ala
435 440 445
Leu Pro Glu Phe Arg Ser Gln Glu Phe Gln Asp Thr Met Val Arg Asn
450 455 460
Ile Cys Gln Ser Val Ala Gln Thr Asn Arg Glu Ile Arg Glu Lys Thr
465 470 475 480
Tyr Arg Thr Phe Asp Leu Val His Arg Gly Leu Ala Asp Arg Ile Arg
485 490 495
Gln Gly Val Glu Glu Leu Val Ala Glu Ala Phe Val Ile Pro Arg Gly
500 505 510
Ser Ser Ser Ser Leu Arg Leu
515
<210> 42
<211> 520
<212> PRT
<213> Aspergillus fumigatus
<400> 42
Met Leu Ile Glu Arg Gly Leu Leu Ser Ile Met Ala Ser Lys Cys Ser
1 5 10 15
Gly Thr Trp Ser Thr Tyr Lys Thr Tyr Thr Thr Ala Asn Gly Cys Pro
20 25 30
Phe Met Lys Pro Glu Gly Pro Pro Ala Asp Gly Arg Thr Leu Ala Leu
35 40 45
Asn Asp His His Leu Val Glu Ser Leu Ala His Phe Asn Arg Glu Lys
50 55 60
Ile Pro Glu Arg Ala Val His Ala Lys Gly Ala Ala Ala Tyr Gly Glu
65 70 75 80
Phe Glu Val Thr Ala Asp Ile Ser Asp Ile Cys Asn Ile Asp Met Leu
85 90 95
Leu Gly Val Gly Lys Lys Thr Pro Cys Val Thr Arg Phe Ser Thr Thr
100 105 110
Gly Leu Glu Arg Gly Ser Ala Glu Gly Met Arg Asp Leu Lys Gly Met
115 120 125
Ala Thr Lys Phe Tyr Thr Lys Glu Gly Asn Trp Asp Trp Val Cys Leu
130 135 140
Asn Phe Pro Phe Phe Phe Ile Arg Asp Pro Leu Lys Phe Pro Ser Leu
145 150 155 160
Met His Ala Gln Arg Arg Asp Pro Arg Thr Asn Leu Leu Asn Pro Asn
165 170 175
Met Tyr Trp Asp Trp Val Thr Ser Asn His Glu Ser Leu His Met Val
180 185 190
Leu Leu Gln Phe Ser Asp Phe Gly Thr Met Phe Asn Trp Arg Ser Leu
195 200 205
Ser Gly Tyr Met Gly His Ala Tyr Lys Trp Val Met Pro Asn Gly Ser
210 215 220
Phe Lys Tyr Val His Ile Phe Leu Ser Ser Asp Arg Gly Pro Asn Phe
225 230 235 240
Ser Gln Gly Glu Gln Ala Lys Asp Asn Ser Asp Leu Asp Pro Asp His
245 250 255
Ala Thr Arg Asp Leu Tyr Glu Ala Ile Glu Arg Gly Asp Tyr Pro Thr
260 265 270
Trp Thr Ala Asn Val Gln Val Val Asp Pro Ala Glu Ala Pro Asp Leu
275 280 285
Gly Phe Asn Ile Leu Asp Val Thr Lys His Trp Asn Leu Gly Thr Tyr
290 295 300
Pro Lys Asp Leu Pro Lys Ile Pro Ser Arg Pro Phe Gly Lys Leu Thr
305 310 315 320
Leu Asn Arg Ile Pro Asp Asn Phe Phe Ala Glu Val Glu Gln Leu Ala
325 330 335
Phe Ser Pro Ser Asn Met Val Pro Gly Val Leu Pro Ser Glu Asp Pro
340 345 350
Ile Leu Gln Ala Arg Met Phe Ala Tyr Pro Asp Ala Gln Arg Tyr Arg
355 360 365
Leu Gly Pro Asn His His Lys Ile Pro Val Asn Gln Cys Pro Met Thr
370 375 380
Phe Asn Pro Thr Leu Arg Asp Gly Thr Gly Thr Phe Asp Ala Asn Tyr
385 390 395 400
Gly Ser Leu Pro Gly Tyr Val Ser Glu Ser Gln Gly Val Asn Phe Ala
405 410 415
Arg Pro Gln Glu His Asp Pro Lys Phe Asn Ala Trp Leu Ser Gln Leu
420 425 430
Ser Ser Arg Pro Trp Met Gln Thr Asn Glu Asn Asp Tyr Lys Phe Pro
435 440 445
Arg Asp Phe Tyr Asn Ala Leu Pro Glu Phe Arg Ser Gln Glu Phe Gln
450 455 460
Asp Lys Met Val Glu Asn Ile Ile Ala Ser Val Ala Gln Thr Arg Arg
465 470 475 480
Glu Ile Arg Glu Lys Val Tyr His Thr Phe His Leu Val Asp Pro Glu
485 490 495
Leu Ser Ala Arg Val Lys Arg Gly Val Glu Lys Met Asp Ala Ser Phe
500 505 510
Lys Gln Val Ser Leu Ser Arg Leu
515 520
<210> 43
<211> 483
<212> PRT
<213> Trichophyton rubrum
<400> 43
Met Ala Pro Asn Ala Ala Asp Lys Cys Pro Val Met Asn Thr Thr Gly
1 5 10 15
Glu Lys Cys Pro Val Met Asn Pro Asn Gly Leu Ser Ser Ser Thr Gln
20 25 30
Ser Arg Gly Pro Arg Asp Ile Tyr Thr Leu Glu Ala Leu Ser His Phe
35 40 45
Asn Arg Glu Lys Ile Pro Glu Arg Ala Val His Ala Lys Gly Thr Gly
50 55 60
Ala Tyr Gly Glu Phe Glu Val Thr Ala Asp Ile Ser Asp Ile Cys Asn
65 70 75 80
Ile Asp Met Leu Leu Gly Val Gly Lys Lys Thr Gln Cys Val Thr Arg
85 90 95
Phe Ser Thr Thr Gly Leu Glu Arg Gly Ser Ser Asp Gly Val Arg Asp
100 105 110
Leu Lys Gly Met Ala Val Lys Phe Phe Thr Glu Gln Gly Asp Trp Asp
115 120 125
Trp Val Ser Leu Asn Phe Pro Phe Phe Phe Ile Arg Asp Pro Ala Lys
130 135 140
Phe Pro Asp Met Ile His Ser Gln Arg Arg Asp Pro Gln Thr Asn Leu
145 150 155 160
Leu Asn Pro Asn Met Thr Trp Asp Phe Val Thr Lys Asn Pro Glu Ala
165 170 175
Leu His Met Thr Leu Leu Gln His Ser Asp Phe Gly Thr Met Phe Thr
180 185 190
Trp Arg Thr Leu Ser Ser Tyr Val Gly His Ala Phe Lys Trp Val Met
195 200 205
Pro Asp Gly Ser Phe Lys Tyr Val His Phe Phe Leu Ala Ser Asp Arg
210 215 220
Gly Pro Asn Phe Thr Asp Gly Ser Thr Ala Lys Ile Asp Pro Asn Asp
225 230 235 240
Pro Asp Phe Ala Thr Lys Asp Leu Phe Glu Ala Ile Glu Arg Gly Asp
245 250 255
Tyr Pro Ser Trp Thr Ala Asn Val Gln Val Val Asp Pro Lys Asp Ala
260 265 270
Pro Lys Leu Gly Phe Asn Ile Leu Asp Ile Thr Lys His Trp Asn Leu
275 280 285
Gly Thr Tyr Pro Lys Gly Leu Asp Thr Ile Pro Ser Arg Pro Phe Gly
290 295 300
Lys Leu Thr Leu Asn Arg Asn Val Lys Asp Tyr Phe Ser Glu Val Glu
305 310 315 320
Lys Leu Ala Phe Ser Pro Ser Asn Leu Val Pro Gly Val Glu Pro Ser
325 330 335
Glu Asp Pro Ile Leu Gln Ala Arg Met Phe Ala Tyr Pro Asp Ala Gln
340 345 350
Arg Tyr Arg Leu Gly Ile Asp His Leu Lys Ala Pro Leu Arg Arg Lys
355 360 365
Glu Thr Ala Cys Gln Gln Asp Leu Gly Pro Glu Phe Glu Lys Trp Leu
370 375 380
Ser Gln Val Thr Ser Glu Ala Trp Ser His Pro Tyr Glu Asp Asp Tyr
385 390 395 400
Lys Phe Ala Arg Glu Tyr Tyr Glu Val Leu Pro Glu Phe Arg Ser Gln
405 410 415
Glu Phe Gln Asp Arg Met Val Glu Asn Leu Cys Arg Ser Ile Ala Pro
420 425 430
Gly Pro Glu Glu Leu Arg Lys Arg Val Phe Asp Thr Phe Glu Leu Val
435 440 445
Ser Ser Glu Leu Ala Arg Arg Leu Arg Glu Gly Val Glu Ala Ile Val
450 455 460
Ala Glu Lys Ala Arg Pro Asp Ser Pro Ser Arg Ala Gln Pro Gly Gln
465 470 475 480
Leu Arg Leu
<210> 44
<211> 482
<212> PRT
<213> Arthroderma otae
<400> 44
Met Ala Pro Asn Ala Ala Asp Lys Cys Pro Val Met Asn Asn Ala Ala
1 5 10 15
Glu Lys Cys Pro Val Met His Ser Asn Gly Val Ser Ser Ile Gln Ser
20 25 30
His Gly Pro Arg Asp Ile Tyr Thr Leu Glu Ala Leu Ser His Phe Asn
35 40 45
Arg Glu Lys Ile Pro Glu Arg Ala Val His Ala Lys Gly Thr Gly Ala
50 55 60
Tyr Gly Glu Phe Glu Val Thr Ala Asp Ile Ser Asp Ile Cys Asn Val
65 70 75 80
Asp Met Leu Leu Gly Val Gly Lys Lys Thr Gln Cys Val Thr Arg Phe
85 90 95
Ser Thr Thr Gly Leu Glu Arg Gly Ser Ser Asp Gly Val Arg Asp Leu
100 105 110
Lys Gly Met Ala Val Lys Phe Phe Thr Glu Gln Gly Asp Trp Asp Trp
115 120 125
Val Ser Leu Asn Phe Pro Phe Phe Phe Ile Arg Asp Pro Ala Lys Phe
130 135 140
Pro Asp Met Ile His Ser Gln Arg Arg Asp Pro Gln Thr Asn Leu Leu
145 150 155 160
Asn Pro Ser Met Thr Trp Asp Phe Val Thr Lys Asn Pro Glu Ala Leu
165 170 175
His Met Thr Leu Leu Gln His Ser Asp Phe Gly Thr Met Phe Thr Trp
180 185 190
Arg Thr Leu Ser Ser Tyr Ala Gly His Ala Phe Lys Trp Val Met Pro
195 200 205
Asp Gly Ser Phe Lys Tyr Val His Phe Phe Leu Ala Ser Asp Arg Gly
210 215 220
Pro Asn Phe Thr Asp Gly Ser Thr Ala Lys Ile Asp Pro Asn Asp Pro
225 230 235 240
Asp Phe Ala Thr Lys Asp Leu Phe Glu Ala Ile Glu Arg Gly Asp Tyr
245 250 255
Pro Thr Trp Thr Ala Asn Val Gln Val Ile Asp Pro Lys Asp Ala Pro
260 265 270
Lys Leu Gly Phe Asn Ile Leu Asp Ile Thr Lys His Trp Asn Leu Gly
275 280 285
Thr Tyr Pro Lys Gly Leu Asp Thr Ile Pro Ser Arg Pro Phe Gly Lys
290 295 300
Leu Thr Leu Asn Arg Asn Val Lys Asp Tyr Phe Thr Glu Val Glu Lys
305 310 315 320
Leu Ala Phe Ser Pro Ser Asn Leu Val Pro Gly Val Glu Pro Ser Glu
325 330 335
Asp Pro Ile Leu Gln Ala Arg Met Phe Ala Tyr Pro Asp Ala Gln Arg
340 345 350
Tyr Arg Leu Gly Ile Asp His Leu Lys Ala Pro Ile Arg Arg Lys Glu
355 360 365
Thr Ser Cys Lys His Asp Leu Gly Pro Glu Phe Asp Gln Trp Leu Ser
370 375 380
Gln Val Thr Ser Glu Ser Trp Ser His Pro His Glu Asp Asp Tyr Lys
385 390 395 400
Phe Ala Arg Glu Tyr Tyr Glu Val Leu Pro Glu Phe Arg Ser Gln Glu
405 410 415
Phe Gln Asp Arg Met Val Glu Asn Leu Tyr Lys Ser Val Ala Gln Gly
420 425 430
Pro Glu Asp Leu Arg Lys Arg Val Tyr Glu Thr Phe Glu Leu Val Ser
435 440 445
Thr Glu Leu Ala Arg Arg Val Arg Glu Gly Ala Glu Val Ile Val Ala
450 455 460
Asp Met Ala Gln Pro Asp Ser Pro Glu Arg Ala Gln Pro Gly Gln Gln
465 470 475 480
Arg Leu
<210> 45
<211> 476
<212> PRT
<213> Periglandula ipomoeae
<400> 45
Met Ala Ser Lys Thr Ser Val Glu Thr Asn Glu Ser Gly Asp Ser Arg
1 5 10 15
Ser Gln Arg Gly Arg Gly Tyr Gln Ala Tyr Ser Ala Ile Asp His Arg
20 25 30
Leu Leu Asp Ser Leu Ser Arg Phe Asn Arg Glu Lys Ile Pro Glu Arg
35 40 45
Ala Val His Ala Arg Gly Ala Gly Ala Tyr Gly Glu Phe Glu Val Thr
50 55 60
Asp Asp Ile Ser Asp Ile Cys Asp Ile Asp Met Leu Leu Gly Ile Gly
65 70 75 80
Lys Lys Thr Pro Cys Val Val Arg Phe Ser Thr Thr Thr Leu Glu Arg
85 90 95
Gly Ser Ala Glu Ala Val Arg Asp Val Lys Gly Met Ala Ile Lys Leu
100 105 110
Phe Thr Gln Asp Gly Asn Trp Asp Trp Val Cys Leu Asn Ile Pro Met
115 120 125
Phe Phe Ile Arg Asp Pro Ser Lys Phe Pro Asp Leu Ile His Ala Gln
130 135 140
Arg Pro Asp Pro Lys Thr Asn Leu Thr Asn Pro Ser Lys Trp Trp Glu
145 150 155 160
Phe Val Cys Asn Asn His Glu Thr Leu His Met Val Met Phe Gln Phe
165 170 175
Ser Asp Phe Gly Thr Met Phe Asp Tyr Arg Ser Met Ser Gly Tyr Val
180 185 190
Ser His Ala Tyr Lys Trp Val Met Pro Asp Gly Ser Trp Lys Tyr Val
195 200 205
His Trp Phe Leu Ala Ser Asp Gln Gly Pro Asn Phe Glu Lys Gly Lys
210 215 220
Gln Thr Lys Glu Ile Ala Pro Asn Asp Pro Glu Ser Ala Thr Arg Asp
225 230 235 240
Leu His Gln Ser Leu Asp Arg Gly Glu Tyr Pro Thr Trp Thr Val Asn
245 250 255
Val Gln Val Val Asp Pro Glu Asp Ala His Gln Leu Ala Phe Asn Ile
260 265 270
Leu Asp Val Thr Lys His Trp Asn Leu Gly Asn Tyr Pro Pro Asp Ile
275 280 285
Pro Val Ile Pro Ser Arg Arg Phe Gly Lys Leu Thr Leu Lys Gln Gly
290 295 300
Pro Lys Asp Tyr Phe Glu Glu Ile Glu Gln Leu Ala Phe Ser Pro Ser
305 310 315 320
His Leu Val His Gly Val Glu Pro Ser Glu Asp Pro Met Leu Gln Ala
325 330 335
Arg Leu Phe Ala Tyr Pro Asp Ala Gln Arg His Arg Leu Gly Pro Gln
340 345 350
Asn Leu Asp Met Pro Ala Asn Arg Thr Arg His Thr Ala Asp Gly Ala
355 360 365
Lys Thr Lys Lys Val Val Pro Pro Glu Lys Lys Ser Arg Glu His Ala
370 375 380
Glu Trp Val Ser Gln Met Thr Ser Ser Ser Trp Ser Gln Pro Asp Glu
385 390 395 400
Ala Asp Tyr Lys Phe Pro Arg Glu Phe Trp Lys Ala Leu Pro Arg Leu
405 410 415
Arg Gly Glu Glu Phe Gln Asn Asn Leu Val Val Asn Met Ser Lys Ser
420 425 430
Val Ser Gln Ile Pro Val Asp Leu Arg Gln Lys Val Tyr Asn Thr Leu
435 440 445
Gly Leu Val Ala Glu Asp Leu Ala Asp Arg Val Lys Ala Met Thr Glu
450 455 460
Glu Met Ile Met Glu Ser Glu Gln Arg Ala Arg Leu
465 470 475
<210> 46
<211> 476
<212> PRT
<213> Epichloe festucae
<400> 46
Met Ser Asp Ile Val Ser Val Ser Val Asn Gly Leu Gln Asn Val Ser
1 5 10 15
Leu Lys Glu Asn Asp Glu Leu Lys Asn Pro Ser Arg Met Asp Phe Arg
20 25 30
Leu Ile Asn Thr Leu Ser Gln Phe Asn Arg Glu Lys Ile Pro Glu Arg
35 40 45
Ala Val His Ala Arg Gly Ala Gly Ala Tyr Gly Glu Phe Glu Val Thr
50 55 60
His Asp Ile Ser Asp Ile Cys Asp Ile Asp Met Leu Leu Gly Ile Gly
65 70 75 80
Lys Lys Thr His Cys Ala Val Arg Phe Ser Thr Thr Ala Leu Glu Arg
85 90 95
Gly Ser Pro Glu Tyr Ile Arg Asp Ala Lys Gly Met Ala Val Lys Phe
100 105 110
Phe Thr Gln Glu Gly Asn Trp Asp Trp Val Cys Leu Asn Ile Pro Met
115 120 125
Phe Phe Ile Arg Asp Pro Ile Lys Phe Pro Ala Leu Ile His Ala Gln
130 135 140
Arg Pro Asp Pro Arg Thr Asn Met Thr Asn Pro Arg Asn Trp Trp Glu
145 150 155 160
Phe Val Cys Asp Asn His Glu Ala Leu His Met Val Met Phe Gln Phe
165 170 175
Ser Asp Phe Gly Thr Met Phe Asn Tyr Arg Ser Met Ser Ala Tyr Val
180 185 190
Gly His Ala Tyr Lys Trp Ala Met Pro Asp Gly Ser Trp Lys Tyr Val
195 200 205
His Trp Phe Leu Ser Ser Asp Gln Gly Pro Asn Phe Glu Asn Gly Ser
210 215 220
Gln Ala Asn Asp Ile Ala Pro Asn Asp Leu Glu Ser Ala Thr Arg Asp
225 230 235 240
Leu Tyr Glu Ser Ile Glu Arg Gly Gly Tyr Pro Thr Trp Thr Ala Asn
245 250 255
Val Gln Val Val Asp Pro Glu Asp Ala His Lys Leu Ala Phe Asn Ile
260 265 270
Leu Asp Leu Thr Lys His Trp Asn Leu Gly Asn Tyr Pro Pro Asp Ile
275 280 285
Pro Val Ile Pro Ser Arg Pro Phe Gly Lys Leu Thr Leu Lys Lys Asn
290 295 300
Pro Lys Asp Tyr Phe Gln Glu Ile Glu Gln Leu Ala Phe Ser Pro Ser
305 310 315 320
His Leu Val Pro Gly Val Glu Pro Ser Glu Asp Pro Met Leu Gln Ala
325 330 335
Arg Leu Phe Ala Tyr Pro Asp Ala Gln Arg Tyr Arg Leu Gly Pro His
340 345 350
Asn Leu Gln Leu Ser Pro Asn Gln Thr Arg His Thr Thr Glu Ser Thr
355 360 365
Arg Ser Arg Ile Ser Ala Arg Thr Lys Ser Ala Asp Pro Lys His Thr
370 375 380
Ala Trp Leu Ser Ala Thr Val Ser Glu Ala Trp Ser Gln Pro Asn Glu
385 390 395 400
Leu Asp Tyr Lys Tyr Pro Arg Glu Phe Trp Lys Ala Leu Pro Arg Leu
405 410 415
Arg Ser Glu Glu Phe Gln Ser Asn Val Val Val Asn Met Ala Lys Ser
420 425 430
Val Ser Gln Val Pro His Glu Leu Arg Glu Arg Val Tyr His Thr Leu
435 440 445
Asn Leu Ile Ala Thr Asp Leu Ala Asp Arg Val Lys Gln Thr Thr Glu
450 455 460
Lys Met Val Val Glu Lys Glu Thr Thr Ala Arg Leu
465 470 475
<210> 47
<211> 516
<212> PRT
<213> Hypocrea atroviridis
<400> 47
Met Val His Ser Asp Asn Val Pro Lys Gly Gly Thr Ser Asp Asn Asp
1 5 10 15
Pro Arg Glu Lys Pro Lys Met Phe Thr Thr Ser Asn Gly Cys Pro Ala
20 25 30
Lys Ser Pro Glu Ser Ser Pro Met Ala Ser Asn Met Leu Leu Leu Arg
35 40 45
Asp Phe Gln Leu Ile Asp Leu Leu Ser His Leu Asn Arg Glu Arg Ile
50 55 60
Pro Glu Arg Val Val His Ala Lys Gly Ala Gly Ala Phe Gly Glu Phe
65 70 75 80
Glu Val Thr His Asp Ile Ser Asp Ile Cys Ser Ile Glu Met Leu Asn
85 90 95
Gly Ile Gly Lys Lys Thr Gln Cys Leu Val Arg Phe Ser Thr Thr Gly
100 105 110
Gly Glu Arg Gly Ser Ala Asp Ser Val Arg Asp Ala Arg Gly Phe Ala
115 120 125
Val Lys Cys Tyr Thr Lys Glu Gly Asn Trp Asp Trp Val Trp Asn Asp
130 135 140
Val Pro Phe Phe Phe Ile Arg Asp Pro Ile Lys Phe Pro Gly Met Ile
145 150 155 160
His Ala Gln Lys Arg Asp Pro Arg Ser Asn Leu Arg Asp Pro Thr Arg
165 170 175
Phe Trp Ser Trp Val Ile Gln Asn Pro Glu Ser Leu His Met Val Met
180 185 190
Trp Leu Tyr Ser Asp Tyr Gly Thr Phe Ser Ser Tyr Arg His Met Asn
195 200 205
Gly Tyr Met Gly His Ala His Lys Trp Val Met Pro Asp Gly Ser Phe
210 215 220
Lys Tyr Val His Met Tyr Leu Glu Ala Asp Leu Gly Tyr Lys Asn His
225 230 235 240
Ser Asp Glu Asp Ile Pro His Met Thr Gly Asn Asp Pro Asp His Ala
245 250 255
Ala Arg Asp Leu Tyr Asp Ala Ile Glu Arg Gly Glu Tyr Pro Thr Tyr
260 265 270
Thr Ala Lys Val Gln Val Ile Asp Pro Ala Asp Val Arg Lys Phe Gly
275 280 285
Tyr Asn Ile Leu Asp Met Thr Lys His Trp Asp Met Gly Thr Tyr Pro
290 295 300
Thr Asp Leu Gly Thr Val Gly Ala Arg Val Val Gly Lys Leu Thr Leu
305 310 315 320
Asn Arg Asn Pro Glu Asn His Phe Ala Gln Ile Glu Gln Ala Ala Phe
325 330 335
Ser Pro Ser His Leu Val Pro Gly Ile Glu Pro Ser Glu Asp Pro Met
340 345 350
Leu Gln Ala Arg Phe Phe Ala Tyr Ala Asp Ala Gln Arg Tyr Arg Leu
355 360 365
Gly Val Asn Tyr Gln Gln Ile Pro Val Asn Gln Pro Lys His Ala Phe
370 375 380
Asn Pro Leu Leu Arg Asp Gly Ala Ala Ser Val Leu Gly Asn Tyr Gly
385 390 395 400
Ser His Pro Gly Phe Val Thr Arg Lys Asp Thr Met Gln Phe Arg His
405 410 415
Phe Glu Ser Ser Ser Thr Pro Glu His Lys Lys Trp Leu Ala Glu Ile
420 425 430
Ala Ser Glu Pro Asn Glu Arg Lys Glu Glu Leu Asp Leu Arg Phe Ala
435 440 445
His Thr Phe Trp Thr His Leu Asp Asp Pro Gln Tyr Glu Gly Trp Gln
450 455 460
Glu Arg Met Val Arg Ser Leu Gly Arg Asp Ile Ala Gly Val Glu Met
465 470 475 480
Glu Thr Arg Asp Gln Val Phe Gln Val Leu Lys Lys Val His Ser Glu
485 490 495
Leu Ala Ala Arg Val Glu Glu Lys Val Glu Glu Arg Leu Ala Gln Ala
500 505 510
Lys Ser Arg Leu
515
<210> 48
<211> 476
<212> PRT
<213> Metarhizium robertsii
<400> 48
Met Ala His Asn Ser Ser Thr Glu Thr Lys Gly Ser His Asp Ala Gly
1 5 10 15
Leu Lys Asp Asn Arg Gly Pro Gln Ser Asn Pro Gly Leu Asn Tyr Asn
20 25 30
Leu Leu Asp Lys Leu Ala Gln Phe Asn Arg Glu Lys Ile Pro Glu Arg
35 40 45
Ala Val His Ala Arg Gly Ala Gly Ala Tyr Gly Glu Phe Glu Val Thr
50 55 60
Asp Asp Ile Ser Asp Ile Cys Asp Ile Asp Met Leu Leu Gly Val Gly
65 70 75 80
Lys Lys Thr Pro Cys Val Val Arg Phe Ser Thr Thr Ala Leu Glu Arg
85 90 95
Gly Ser Ala Glu Ala Val Arg Asp Val Lys Gly Leu Ala Val Lys Phe
100 105 110
Phe Thr Gln Gln Gly Asn Trp Asp Trp Val Cys Leu Asn Ile Pro Met
115 120 125
Phe Phe Ile Arg Asp Pro Ile Lys Phe Pro Asp Leu Val His Ala Gln
130 135 140
Arg Pro Asp Pro Lys Thr Asn Leu Thr Asn Pro Ser Asn Trp Trp Glu
145 150 155 160
Phe Val Cys Asn Asn His Glu Thr Leu His Met Val Met Phe Gln Phe
165 170 175
Ser Asp Phe Gly Thr Met Phe Asp Tyr Arg Ser Met Ser Gly Tyr Val
180 185 190
Gly His Ala Tyr Lys Trp Val Lys Pro Asp Gly Ser Trp Lys Tyr Val
195 200 205
His Trp Phe Leu Ser Ser Asp Gln Gly Pro Asn Phe Glu Gln Gly Ser
210 215 220
Gln Thr Lys Glu Leu Ala Pro Asp Asp Gln Glu Ser Ala Thr Arg Asp
225 230 235 240
Leu Asn Glu Ser Leu Glu Arg Gly Glu Cys Pro Ser Trp Thr Ala Asn
245 250 255
Val Gln Val Ile Asp Pro Glu Asp Ala His Lys Leu Pro Phe Asn Ile
260 265 270
Leu Asp Val Thr Lys His Trp Asn Leu Gly Asn Tyr Pro Pro Asp Ile
275 280 285
Pro Val Ile Pro Ser Arg Arg Phe Gly Lys Leu Thr Leu Lys Lys Gly
290 295 300
Pro Lys Asp Tyr Phe Gln Glu Ile Glu Gln Leu Ala Phe Ser Pro Ser
305 310 315 320
His Leu Val Pro Gly Val Glu Pro Ser Glu Asp Pro Met Leu Gln Ala
325 330 335
Arg Leu Phe Ala Tyr Pro Asp Ala Gln Arg His Arg Leu Gly Pro His
340 345 350
Asn Leu Glu Met Pro Ala Asn Gln Pro Arg Gln Thr Val Glu Asp Thr
355 360 365
Lys Ala Lys Asp Ser Val Arg Pro Glu Pro Lys Ser Gln Glu His Ser
370 375 380
Ala Trp Val Ser Glu Thr Thr Ser Ser Glu Trp Ser Gln Pro Asn Glu
385 390 395 400
Leu Asp Tyr Lys Phe Pro Arg Glu Phe Trp Lys Ser Leu Pro Val Leu
405 410 415
Arg Asp Asp Glu Phe Gln Asn Asn Val Val Val Asn Ile Ala Glu Ser
420 425 430
Val Ser Arg Thr Pro Ser Ala Leu Arg Arg Lys Val Tyr Arg Thr Leu
435 440 445
Gly Leu Val Ala Ser Asp Leu Ala Glu Arg Val Glu Lys Met Thr Glu
450 455 460
Glu Leu Val Met Glu Val Glu Lys Lys Ala Lys Leu
465 470 475
<210> 49
<211> 473
<212> PRT
<213> Claviceps purpurea
<400> 49
Met Ala Ser Glu Val Ser Val Ala Ser Ser Gly Ser Glu His Ser Gly
1 5 10 15
Ala Gln Lys Cys Pro Phe Gln Asp Pro Gly Leu Ser Ser Met Asp Gln
20 25 30
Asp Ser Arg Leu Arg Asp Ile Leu Ser Arg Phe Asn Arg Glu Lys Ile
35 40 45
Pro Glu Arg Ala Val His Ala Arg Gly Ala Gly Ala Tyr Gly Glu Phe
50 55 60
Glu Val Thr His Asp Val Ser Asp Ile Cys Asp Ile Asp Met Leu Leu
65 70 75 80
Gly Val Gly Lys Lys Thr Pro Cys Val Val Arg Phe Ser Thr Thr Thr
85 90 95
Leu Glu Arg Gly Ser Ala Glu Ser Val Arg Asp Val Lys Gly Met Ala
100 105 110
Ile Lys His Phe Thr Gln Asp Gly Asn Trp Asp Trp Val Cys Leu Asn
115 120 125
Ile Pro Met Phe Phe Ile Arg Asp Pro Ser Lys Phe Pro Asp Met Val
130 135 140
His Ala Gln Arg Pro Asp Pro Thr Thr Asn Val Ala Asn Pro Ser Arg
145 150 155 160
Trp Trp Glu Phe Val Cys Asn Asn His Glu Thr Leu His Met Val Met
165 170 175
Phe Gln Phe Ser Asp Phe Gly Thr Met Phe Asp Tyr Arg Ser Met Ser
180 185 190
Gly Tyr Ala Ala His Ala Tyr Lys Trp Val Met Pro Asp Gly Ser Trp
195 200 205
Lys Tyr Val His Trp Phe Leu Ala Ser Asp Gln Gly Pro Asn Phe Glu
210 215 220
Thr Gly His Gln Ala Lys Gln Ile Gly Ala Asp Asp Ala Glu Ser Ala
225 230 235 240
Thr Arg Asp Leu Tyr Gln Ser Leu Glu Arg Gly Glu Tyr Pro Ser Trp
245 250 255
Thr Val Lys Val Gln Val Val Asp Pro Glu Asp Ala Pro Lys Leu Pro
260 265 270
Phe Asn Ile Leu Asp Val Thr Lys His Trp Asn Leu Gly Asn Tyr Pro
275 280 285
Pro Asp Ile Asp Val Ile Pro Gly Arg Thr Leu Gly Lys Leu Thr Leu
290 295 300
Lys Lys Gly Pro Gln Asp Tyr Phe Glu Glu Ile Glu Gln Leu Ala Phe
305 310 315 320
Ser Pro Ser Arg Leu Val His Gly Val Glu Ala Ser Glu Asp Pro Met
325 330 335
Leu Gln Ala Arg Leu Phe Ala Tyr Pro Asp Ala Gln Lys His Arg Leu
340 345 350
Gly Pro Asn Asn Leu Asp Leu Pro Ala Asn Arg Thr Lys Lys Leu Ala
355 360 365
Asp Gly Ser Arg Pro Glu Lys Ala Glu Met Ala Pro Gln Lys Val Pro
370 375 380
Ser Gln Glu His Ala Asp Trp Val Ser Gln Val Lys Ser Ser Ser Trp
385 390 395 400
Ser Glu Pro Asn Glu Thr Asp Tyr Lys Phe Pro Arg Glu Phe Trp Lys
405 410 415
Ala Leu Pro Arg Leu Arg Gly Glu Ala Phe Gln Asn Ser Leu Val Val
420 425 430
Asn Met Ala Lys Ser Val Ser Gln Val Pro Ala Asp Met Arg Gln Lys
435 440 445
Val Tyr Ser Thr Leu Ala Leu Ile Ala Asp Asp Leu Ala Asp Arg Val
450 455 460
Arg Thr Met Thr Glu Glu Ile Val Glu
465 470
<210> 50
<211> 500
<212> PRT
<213> Aspergillus terreus
<400> 50
Met Ala Lys Asp Asp Glu Pro Lys Thr Tyr Arg Tyr Asn Glu Thr Pro
1 5 10 15
Thr Tyr Thr Thr Ser Asn Gly Cys Pro Val Phe Asp Pro Gln Ala Ala
20 25 30
Gln Arg Ile Gly Arg Asn Gly Pro Leu Leu Leu Gln Asp Phe His Leu
35 40 45
Ile Asp Leu Leu Ala His Phe Asp Arg Glu Arg Ile Pro Glu Arg Val
50 55 60
Val His Ala Lys Gly Ala Gly Ala Tyr Gly Glu Phe Glu Val Thr Asp
65 70 75 80
Asp Ile Ser Asp Ile Thr Thr Ile Asp Met Leu Lys Gly Val Gly Lys
85 90 95
Lys Thr Lys Thr Phe Val Arg Phe Ser Thr Val Gly Gly Glu Lys Gly
100 105 110
Ser Pro Asp Ser Ala Arg Asp Pro Arg Gly Phe Ala Val Lys Phe Tyr
115 120 125
Thr Glu Gln Gly Asn Trp Asp Trp Val Phe Asn Asn Thr Pro Val Phe
130 135 140
Phe Leu Arg Asp Pro Ala Lys Phe Pro Ile Phe Ile His Thr Gln Lys
145 150 155 160
Arg Asn Pro Gln Thr Asn Leu Lys Asp Ala Thr Met Phe Trp Asp Tyr
165 170 175
Leu Ser Thr His His Glu Ala Val His Gln Ile Met His Leu Phe Ser
180 185 190
Asp Arg Gly Thr Pro Tyr Ser Tyr Arg His Met Asn Gly Tyr Ser Gly
195 200 205
His Thr Tyr Lys Trp Ile Lys Pro Asp Gly Thr Phe Asn Tyr Val Gln
210 215 220
Ile His Leu Lys Thr Asp Gln Gly Asn Lys Thr Phe Asn Asn Glu Glu
225 230 235 240
Ala Thr Arg Leu Ala Ala Glu Asn Pro Asp Trp His Thr Glu Asp Leu
245 250 255
Phe Asn Ala Ile Glu Arg Gly Glu Tyr Pro Ser Trp Thr Cys Tyr Val
260 265 270
Gln Val Leu Ser Pro Glu Gln Ala Glu Lys Phe Arg Trp Asn Val Phe
275 280 285
Asp Leu Thr Lys Val Trp Pro Gln Ser Glu Val Pro Leu Arg Arg Phe
290 295 300
Gly Arg Phe Thr Leu Asn Lys Asn Pro Gln Asn Tyr Phe Ala Glu Val
305 310 315 320
Glu Gln Ala Ala Phe Ser Pro Ser His Met Val Pro Gly Val Glu Pro
325 330 335
Ser Ala Asp Pro Val Leu Gln Ser Arg Leu Phe Ser Tyr Pro Asp Thr
340 345 350
His Arg His Arg Leu Gly Gly Asn Tyr Glu Gln Ile Pro Val Asn Cys
355 360 365
Pro Leu Arg Ala Phe Asn Pro Thr His Arg Asp Gly Tyr Met Asn Val
370 375 380
His Gly Asn Tyr Gly Ala Asn Pro Asn Tyr Pro Ser Thr Phe Arg Pro
385 390 395 400
Ile Arg Tyr Glu Pro Val Lys Ala Ser Gln Glu His Glu Lys Trp Ala
405 410 415
Gly Ser Val Val Val Glu Gln Leu Pro Val Thr Ser Glu Asp Tyr Val
420 425 430
Gln Ala Asn Gly Leu Trp Gln Val Leu Gly Arg Gln Pro Gly Gln Gln
435 440 445
Asp Ala Phe Val His Asn Val Ser Val His Leu Cys Gly Ala His Glu
450 455 460
Arg Val Arg Lys Ala Thr Tyr Asp Met Phe Arg Arg Val Asn Pro Asp
465 470 475 480
Leu Gly Ala Arg Ile Glu Lys Ala Thr Glu Ala Asn Val Ser Ala Ala
485 490 495
Arg Ala Arg Leu
500
<210> 51
<211> 500
<212> PRT
<213> Penicillium marneffei
<400> 51
Met Gly Lys Asp Asp Glu Pro Lys Thr Tyr Arg Tyr Asn Glu Thr Pro
1 5 10 15
Thr Tyr Thr Thr Ser Asn Gly Cys Pro Val Phe Asp Pro Glu Ser Ser
20 25 30
Gln Arg Ile Gly Lys Asn Gly Pro Leu Leu Leu Gln Asp Phe His Leu
35 40 45
Ile Asp Leu Leu Ala His Phe Asp Arg Glu Arg Ile Pro Glu Arg Val
50 55 60
Val His Ala Lys Gly Ala Gly Ala Tyr Gly Glu Phe Glu Val Thr Asp
65 70 75 80
Asp Ile Ser Asp Ile Thr Thr Ile Asp Met Leu Lys Gly Val Gly Lys
85 90 95
Lys Thr Lys Leu Val Thr Arg Phe Ser Thr Val Gly Gly Glu Lys Gly
100 105 110
Ser Ala Asp Ser Ala Arg Asp Pro Arg Gly Phe Ser Val Lys Phe Tyr
115 120 125
Thr Glu Gln Gly Asn Trp Asp Trp Val Phe Asn Asn Thr Pro Val Phe
130 135 140
Phe Leu Arg Asp Pro Ser Lys Phe Pro Val Phe Ile His Thr Gln Lys
145 150 155 160
Arg Asn Pro Gln Thr Asn Leu Lys Asp Ala Asn Met Phe Trp Asp Tyr
165 170 175
Leu Ser Thr His Gln Glu Ser Ala His Gln Val Met His Leu Phe Ser
180 185 190
Asp Arg Gly Thr Pro Tyr Ser Tyr Arg His Met Asn Gly Tyr Ser Gly
195 200 205
His Thr Phe Lys Trp Ile Lys Pro Asp Gly Thr Phe Asn Tyr Val Gln
210 215 220
Ile His Leu Lys Thr Asp Gln Gly Asn Lys Thr Leu Thr Asn Glu Glu
225 230 235 240
Ala Gly Glu Leu Ser Asn Ser Asn Pro Asp Trp His Thr Gln Asp Leu
245 250 255
Phe Gln Ala Ile Glu Arg Gly Glu Tyr Pro Ser Trp Thr Cys Tyr Val
260 265 270
Gln Thr Leu Ser Pro Glu Gln Ala Glu Lys Phe Arg Trp Asn Val Phe
275 280 285
Asp Leu Thr Lys Val Trp Pro Gln Ser Glu Val Pro Leu Arg Arg Phe
290 295 300
Gly Arg Phe Thr Leu Asn Lys Asn Pro Glu Asn Tyr Phe Ala Glu Ile
305 310 315 320
Glu Gln Ala Ala Phe Ala Pro Ser His Leu Val Pro Gly Val Glu Pro
325 330 335
Ser Ala Asp Pro Val Leu Gln Ala Arg Leu Phe Ser Tyr Pro Asp Thr
340 345 350
His Arg His Arg Leu Gly Val Asn Tyr Gln Gln Ile Pro Val Asn Cys
355 360 365
Pro Leu His Ala Phe Asn Pro Tyr Gln Arg Asp Gly Ala Met Ala Val
370 375 380
Asn Gly Asn Tyr Gly Ala Asn Pro Ala Tyr Pro Ser Ser Phe Arg Ala
385 390 395 400
Met Asn Phe Glu Pro Val Lys Ala Ser Gln Glu His Glu Asn Trp Val
405 410 415
Gly Ala Val Ile Ser Gln Gln Ile Pro Val Thr Asp Glu Asp Phe Val
420 425 430
Gln Ala Asn Ala Leu Trp Arg Val Leu Gly Arg Thr Pro Gly Gln Gln
435 440 445
Glu Asn Phe Val Lys Asn Val Ser Val His Leu Cys Asn Ala Asn Glu
450 455 460
Arg Val Arg Lys Gln Thr Tyr Gly Phe Phe Thr Arg Ile Asn Ala Ile
465 470 475 480
Leu Gly Glu Arg Ile Lys Lys Ala Thr Glu Lys Asn Val Ser Arg Glu
485 490 495
His Ala Arg Leu
500
<210> 52
<211> 503
<212> PRT
<213> Ajellomyces dermatitidis
<400> 52
Met Gly Ala Asp Asp Lys Phe Asn Thr Tyr Arg Tyr Asn Glu Thr Pro
1 5 10 15
Thr Tyr Thr Thr Ser Asn Gly Cys Pro Val Met Asp Pro Glu Ser Ser
20 25 30
Gln Arg Val Gly Pro Asp Gly Pro Leu Leu Leu Gln Asp Phe His Leu
35 40 45
Ile Asp Leu Leu Ala His Phe Asp Arg Glu Arg Ile Pro Glu Arg Val
50 55 60
Val His Ala Lys Gly Ala Gly Ala Tyr Gly Glu Phe Glu Val Leu Asp
65 70 75 80
Asp Ile Ser Asp Ile Thr Thr Ile Asp Met Leu Arg Gly Val Gly Lys
85 90 95
Lys Thr Lys Leu Val Thr Arg Phe Ser Thr Val Gly Gly Glu Lys Gly
100 105 110
Ser Ala Asp Ser Ala Arg Asp Pro Arg Gly Phe Ser Thr Lys Phe Tyr
115 120 125
Thr Glu Glu Gly Asn Trp Asp Trp Val Phe Asn Asn Thr Pro Val Phe
130 135 140
Phe Leu Arg Asp Pro Ser Lys Phe Pro Leu Phe Ile His Thr Gln Lys
145 150 155 160
Arg Asn Pro Gln Thr Asn Leu Lys Asp Ala Thr Met Phe Trp Asp Tyr
165 170 175
Leu Ser Thr His Gln Glu Ala Ala His Gln Val Met His Leu Phe Ser
180 185 190
Asp Arg Gly Thr Pro Tyr Ser Tyr Arg His Met Asn Gly Tyr Ser Gly
195 200 205
His Thr Tyr Lys Trp Ile Lys Pro Asp Gly Thr Phe Asn Tyr Val Gln
210 215 220
Ile His Cys Lys Thr Asp Gln Gly Asn Lys Thr Leu Thr Asn Glu Glu
225 230 235 240
Ala Thr Lys Leu Ser Ala Asp Asn Pro Asp Trp His Thr Glu Asp Leu
245 250 255
Phe Arg Ala Ile Glu Arg Gly Glu Tyr Pro Ser Trp Thr Cys Tyr Val
260 265 270
Gln Val Leu Ser Pro Glu Gln Ala Glu Lys Phe Arg Trp Asn Ile Phe
275 280 285
Asp Leu Thr Lys Val Trp Pro Gln Gly Asp Val Pro Leu Arg Arg Phe
290 295 300
Gly Arg Phe Thr Leu Asn Lys Asn Pro Gln Asn Tyr Phe Ala Glu Ile
305 310 315 320
Glu Gln Ala Ala Phe Ser Pro Ser His Leu Val Pro Gly Val Glu Pro
325 330 335
Ser Ala Asp Pro Val Leu Gln Ser Arg Leu Phe Ser Tyr Pro Asp Thr
340 345 350
His Arg His Arg Leu Gly Val Asn Tyr Gln Gln Ile Pro Val Asn Cys
355 360 365
Pro Leu Arg Ala Phe Ser Pro Tyr Gln Arg Asp Gly Ala Met Ala Val
370 375 380
Asn Gly Asn Tyr Gly Ala Asn Pro Asn Tyr Pro Ser Thr Phe Arg Pro
385 390 395 400
Leu Ala Phe Ala Pro Val Lys Ala Ser Gln Glu His Glu Gln Trp Ala
405 410 415
Gly Ala Ala Val Ser Lys Gln Phe Pro Val Thr Asp Glu Asp Phe Val
420 425 430
Gln Pro Asn Gly Leu Trp Gln Val Leu Gly Arg Gln Pro Gly Gln Gln
435 440 445
Asp Asn Phe Val Lys Asn Val Ala Ala His Leu Cys Asn Ala Gln Gln
450 455 460
Arg Val Arg Lys Ala Thr Tyr Gly Met Phe Thr Arg Ile Asn Val Asp
465 470 475 480
Leu Gly Asn Arg Ile Glu Lys Ala Thr Glu Ala Leu Val Ala Ala Gln
485 490 495
Gly Gln Ala Gln Ser Arg Leu
500
<210> 53
<211> 261
<212> PRT
<213> Paecilomyces divaricatus
<400> 53
Met Ala Ser Val Ser Ser Arg Ile Phe Ala Ile Thr Gly Gly Ala Ser
1 5 10 15
Gly Ile Gly Ala Ala Thr Cys Arg Leu Leu Ala Gln Arg Gly Ala Ala
20 25 30
Val Ile Cys Val Gly Asp Ile Ser His Gly Asn Phe Asp Asp Leu Lys
35 40 45
Lys Ser Ile Lys Glu Ile Asn Pro Ser Thr Glu Val His Cys Thr Ser
50 55 60
Leu Asp Val Thr Ser Val Ala Glu Val Asp Lys Trp Ile Gln Ser Ile
65 70 75 80
Ile Ser Ser Phe Gly Asp Leu His Gly Ala Ala Asn Val Ala Gly Ile
85 90 95
Ala Gln Gly Ala Gly Met Arg Lys Ser Pro Thr Ile Val Glu Glu Thr
100 105 110
Asp Glu Glu Trp Asn Lys Ile Phe Lys Val Asn Leu Asp Gly Val Phe
115 120 125
Tyr Cys Thr Arg Ala Glu Val Arg Ala Met Lys Asp Leu Pro Arg Gly
130 135 140
Ser Arg Ser Ile Val Asn Val Gly Ser Ile Ala Ala Leu Ser His Met
145 150 155 160
Pro Asp Val Tyr Ala Tyr Gly Thr Ser Lys Gly Ala Ala Ala Tyr Phe
165 170 175
Thr Thr Cys Ala Ala Ala Asp Ser Phe Pro Leu Gly Ile Arg Ile Asn
180 185 190
Gly Val Ser Pro Gly Ile Thr Asp Thr Gln Leu Leu Pro Gln Phe Met
195 200 205
Pro Gly Ala Lys Ser Ala Asp Glu Val Lys Glu Ala Tyr Glu Lys Glu
210 215 220
Gly Phe Thr Ile Ile Lys Pro Asp Asp Val Ala Arg Thr Ile Val Trp
225 230 235 240
Leu Leu Ser Glu Asp Ser Thr Pro Val Tyr Gly Ala Asn Ile Asn Val
245 250 255
Gly Ala Gly Val Pro
260
<210> 54
<211> 261
<212> PRT
<213> Aspergillus fumigatus Af293
<400> 54
Met Ala Ser Val Glu Ser Arg Ile Ile Ala Ile Thr Gly Gly Ala Ser
1 5 10 15
Gly Ile Gly Ala Ala Thr Cys Arg Leu Leu Ala Glu Arg Gly Ala Ala
20 25 30
Val Leu Cys Val Cys Asp Ile Ser Pro Lys Asn Phe Asp Asp Leu Lys
35 40 45
Ile Ser Ile Lys Lys Ile Asn Pro Ser Thr Lys Val His Cys Ala Thr
50 55 60
Val Asp Val Thr Ser Ser Val Glu Val Arg Gln Trp Ile Glu Gly Ile
65 70 75 80
Ile Ser Asp Phe Gly Asp Leu His Gly Ala Val Asn Ala Ala Gly Ile
85 90 95
Ala Gln Gly Ala Gly Met Arg Asn Thr Pro Thr Ile Ala Glu Glu Val
100 105 110
Asp Glu Glu Trp Thr Arg Ile Met Asn Thr Asn Leu Asn Gly Val Phe
115 120 125
Tyr Cys Thr Arg Glu Glu Val Arg Ala Met Lys Gly Leu Pro Ala Thr
130 135 140
Asp Arg Ser Ile Val Asn Val Gly Ser Ile Ala Ser Val Ser His Met
145 150 155 160
Pro Asp Val Tyr Ala Tyr Gly Thr Ser Lys Gly Ala Cys Ala Tyr Phe
165 170 175
Thr Thr Cys Val Ala Ala Asp Ala Phe Pro Leu Gly Ile Arg Ile Asn
180 185 190
Asn Val Ser Pro Gly Val Thr Asn Thr Pro Met Leu Pro Gln Phe Ala
195 200 205
Pro Met Ala Lys Thr Phe Glu Glu Ile Glu Glu Ser Tyr Lys Lys Glu
210 215 220
Gly Leu Ser Leu Ile Glu Ala Glu Asp Val Ala Arg Thr Ile Val Trp
225 230 235 240
Leu Leu Ser Glu Asp Ser Arg Pro Val Phe Gly Ala Asn Ile Asn Val
245 250 255
Gly Ala Cys Met Pro
260
<210> 55
<211> 264
<212> PRT
<213> Arthroderma gypseum
<400> 55
Met Ala Ser Val Thr Ser Lys Ile Phe Ala Ile Thr Gly Gly Ala Ser
1 5 10 15
Gly Ile Gly Ala Ala Thr Cys Arg Leu Leu Ala Gln Arg Glu Ala Ala
20 25 30
Ala Leu Cys Leu Gly Asp Leu Ser Ser Glu Asn Met Lys Gln Leu Glu
35 40 45
Arg Glu Ile Lys Glu Ile Asn Pro Glu Thr Lys Val His Cys Thr Val
50 55 60
Leu Asp Val Ser Ser Ser Ser Glu Val Asp Glu Trp Val Lys Ala Ile
65 70 75 80
Ile Thr Thr Phe Gly Glu Leu His Gly Ala Ala Asn Ile Ala Gly Ile
85 90 95
Ala Gln Gly Ala Gly Leu Arg Gln Thr Pro Thr Leu Leu Glu Glu Asp
100 105 110
Asp Glu Gln Trp Lys Lys Val Phe Arg Val Asn Leu Asp Gly Ile Leu
115 120 125
Tyr Ser Thr Arg Ala Glu Val Arg Ala Met Lys Glu Tyr Ser Leu Thr
130 135 140
Ser Pro Gly Asp Arg Ser Ile Val Asn Val Ala Ser Ile Ala Ser Met
145 150 155 160
Ala His Met Pro Asp Val Phe Ala Tyr Gly Thr Ser Lys Ala Gly Cys
165 170 175
Ala Tyr Phe Thr Thr Cys Val Ala Gln Asp Val Met Pro Phe Gly Ile
180 185 190
Arg Ala Asn Thr Val Ser Pro Gly Ile Thr Arg Thr Pro Met Leu Pro
195 200 205
Lys Phe Val Pro Asn Ala Lys Thr Gln Glu Glu Val Glu Glu Thr Tyr
210 215 220
Lys Lys Glu Gly Tyr Ser Val Ile Glu Ala Asp Asp Val Ala Arg Thr
225 230 235 240
Ile Val Trp Leu Leu Ser Glu Asp Ser Arg Pro Val Phe Gly Ala Asn
245 250 255
Ile Asn Val Gly Ala Cys Met Pro
260
<210> 56
<211> 264
<212> PRT
<213> Trichophyton tonsurans
<400> 56
Met Ala Ser Val Ser Ser Lys Ile Phe Ala Ile Thr Gly Gly Ala Ser
1 5 10 15
Gly Ile Gly Ala Ala Thr Cys Arg Leu Leu Ala Lys Arg Gly Ala Ser
20 25 30
Ala Leu Cys Val Gly Asp Leu Ser Ser Glu Asn Met Lys Gln Leu Glu
35 40 45
Lys Asp Ile Lys Glu Ile Asn Pro Asp Thr Lys Ile His Cys Thr Ile
50 55 60
Leu Asp Val Ser Ser Ser Ser Asn Val Asp Glu Trp Ile Lys Asp Ile
65 70 75 80
Ile Thr Thr Phe Gly Asp Leu His Gly Ala Ala Asn Ile Ala Gly Ile
85 90 95
Ala Gln Gly Ala Gly Leu Arg Gln Ala Pro Thr Ile Leu Glu Glu Asp
100 105 110
Asp Gln Gln Trp Lys Lys Val Phe Gln Val Asn Leu Asp Gly Ile Leu
115 120 125
Tyr Ser Thr Arg Ala Gln Val Arg Ala Met Lys Glu Ser Ser Ser Ile
130 135 140
Asn Pro Gly Asp Arg Ser Ile Val Asn Val Ala Ser Ile Ala Ser Met
145 150 155 160
Ser His Met Pro Asp Val Phe Ala Tyr Gly Thr Ser Lys Ala Gly Cys
165 170 175
Ala Tyr Phe Thr Thr Cys Val Ser Gln Asp Val Met Pro Phe Gly Ile
180 185 190
Arg Ala Asn Thr Val Ser Pro Gly Ile Thr Arg Thr Pro Met Leu Pro
195 200 205
Arg Phe Val Pro Asn Ala Lys Thr Gln Glu Glu Val Glu Glu Thr Tyr
210 215 220
Lys Lys Glu Gly Phe Ser Val Ile Glu Ala Asp Asp Val Ala Arg Thr
225 230 235 240
Ile Val Trp Leu Leu Ser Glu Asp Ser Arg Pro Val Phe Gly Thr Asn
245 250 255
Ile Asn Val Gly Ala Cys Met Pro
260
<210> 57
<211> 262
<212> PRT
<213> Epichloe typhnia
<400> 57
Met Ala Ser Ala Ser Ser Lys Ile Phe Ala Ile Thr Gly Gly Ala Ser
1 5 10 15
Gly Ile Gly Ala Ala Thr Cys Arg Leu Leu Ala Asn Arg Asp Ala Ala
20 25 30
Val Ile Cys Val Ala Asp Val Val Ser Thr Asn Phe Asp Ser Leu Lys
35 40 45
Lys Ser Val His Glu Ile Asn Gln Ser Thr Leu Val Gln Cys Thr Val
50 55 60
Val Asp Val Ala Ser Ser Ser Ala Val Asn Lys Trp Leu Glu Asp Ile
65 70 75 80
Val Ser Glu His Cys Asp Leu His Gly Ala Ala Asn Val Ala Gly Ile
85 90 95
Ala Gln Gly Ala Gly Leu Arg Gln Ser Pro Lys Ile Leu Ala Glu Thr
100 105 110
Asp Glu Asp Trp Ser Arg Ile Met Lys Val Asn Leu Asp Gly Val Phe
115 120 125
Tyr Cys Thr Arg Ala Glu Val Val Ala Met Lys Asp Leu Pro Ala Gly
130 135 140
Asn Arg Ser Ile Val Asn Val Ala Ser Ile Ala Ala Phe Ser His Val
145 150 155 160
Pro Asp Val Phe Ala Tyr Gly Thr Ser Lys Gly Ala Cys Ala Tyr Phe
165 170 175
Thr Ala Cys Val Ala Thr Asp Val Phe Pro Val Gly Ile Arg Val Asn
180 185 190
Cys Val Ser Pro Ala Gly Val Thr Asn Thr Pro Leu Leu Pro Gln Phe
195 200 205
Glu Pro Asn Ala Lys Ser Leu Ala Glu Val Lys Ser Leu Tyr Glu Ser
210 215 220
Gln Gly Phe Ser Ile Val Glu Pro Glu Asp Val Ala Arg Thr Ile Val
225 230 235 240
Trp Leu Leu Ser Glu Asp Ser Trp Pro Val Tyr Gly Ser Asn Ile Asn
245 250 255
Val Gly Ala Ser Ala Pro
260
<210> 58
<211> 261
<212> PRT
<213> Claviceps purpurea
<400> 58
Met Pro Ser Met Thr Ser Lys Val Phe Ala Ile Thr Gly Gly Ala Ser
1 5 10 15
Gly Ile Gly Ala Ala Thr Cys Arg Leu Leu Ala Glu Arg Gly Ala Ala
20 25 30
Val Ile Cys Leu Ala Asp Val Ser Ser Thr Asn Phe Thr Ser Leu Gln
35 40 45
Glu Ser Ile Ala Lys Ser Asn Pro Ser Thr Leu Val His Cys Thr Glu
50 55 60
Leu Asp Val Arg Ser Ala Asp Lys Val Asp Gln Trp Leu Gln Ser Ile
65 70 75 80
Val Ser Thr His Gly Asp Leu His Gly Ala Ala Asn Val Ala Gly Ile
85 90 95
Ala Gln Gly Ala Gly Leu Arg Ala Thr Pro Thr Ile Leu Glu Glu Asn
100 105 110
Asp Ala Glu Trp Ser Arg Ile Leu Asp Val Asn Leu Asn Gly Val Phe
115 120 125
Tyr Ser Thr Arg Ala Gln Val Arg Val Met Lys Asp Leu Pro Pro Gly
130 135 140
His Arg Ser Ile Val Asn Val Ala Ser Ile Ala Ala Phe Ser His Val
145 150 155 160
Pro Asp Val Tyr Ala Tyr Gly Thr Ser Lys Ser Ala Cys Ala Tyr Leu
165 170 175
Thr Thr Cys Ile Ala Ala Asp Val Phe Trp Ser Gly Ile Arg Val Asn
180 185 190
Cys Val Ser Pro Gly Ile Thr Asn Thr Pro Met Leu Pro Gln Phe Glu
195 200 205
Pro Lys Ala Lys Ser Leu Asp Glu Ile Lys Asp Met Tyr Arg Asp Gln
210 215 220
Gly Tyr Pro Thr Gly Glu Ala Asp Gly Val Ala Arg Thr Ile Val Trp
225 230 235 240
Leu Leu Ser Glu Asp Ser Ile Pro Val Tyr Gly Ala Asn Ile Asn Val
245 250 255
Gly Ala Cys Pro Pro
260
<210> 59
<211> 261
<212> PRT
<213> Periglandula ipomoeae
<400> 59
Met Ser Ser Val Ser Ser Lys Ile Phe Ala Ile Thr Gly Gly Ala Ser
1 5 10 15
Gly Ile Gly Ala Ala Thr Cys Arg Leu Leu Ala Asp Arg Gly Ala Ala
20 25 30
Ile Ile Cys Leu Ala Asp Val Ser Ser Asn Asn Phe Val Ser Leu Gln
35 40 45
Glu Ser Ile Ala Glu Val Asn Pro Ser Thr Leu Val Gln Cys Thr Val
50 55 60
Leu Asp Val Ser Ser Ala Val Asp Val Asn Gln Trp Leu Gln Asp Ile
65 70 75 80
Val Ser Ile His Gly Asp Leu His Gly Ala Ala Asn Val Ala Gly Ile
85 90 95
Ala Gln Gly Ala Gly Leu Arg Ala Thr Pro Thr Ile Leu Glu Glu Lys
100 105 110
Asp Ala Asp Trp Ser Arg Ile Leu Asp Val Asn Leu Asn Gly Val Phe
115 120 125
Tyr Ser Thr Arg Ala Gln Val Arg Ala Met Lys Asn Leu Pro Cys Gly
130 135 140
Asn Arg Ser Ile Val Asn Val Ala Ser Ile Ser Ala Phe Ser His Val
145 150 155 160
Pro Asp Val Tyr Ala Tyr Ser Thr Ser Lys Asn Ala Cys Ala Tyr Leu
165 170 175
Thr Thr Cys Ile Ala Ala Asp Val Phe Trp Ser Gly Ile Arg Val Asn
180 185 190
Cys Val Ser Pro Gly Ile Thr Asn Thr Pro Leu Leu Pro Gln Phe Glu
195 200 205
Pro Lys Ala Lys Ser Leu Asn Glu Ile Gln Glu Ile Tyr Arg Arg Gln
210 215 220
Gly Tyr Ser Thr Cys Glu Ala Glu Asp Val Ala Arg Thr Ile Val Trp
225 230 235 240
Leu Leu Ser Asp Asp Ser Arg Pro Val Tyr Gly Ala Asn Ile Asn Val
245 250 255
Gly Ala Cys Pro Pro
260
<210> 60
<211> 261
<212> PRT
<213> Metarhizium robertsii
<400> 60
Met Ala Ser Leu Thr Ala Lys Ile Phe Ala Val Thr Gly Gly Ala Ser
1 5 10 15
Gly Ile Gly Ala Ala Thr Cys Arg Leu Leu Ala Glu Arg Gly Ala Gly
20 25 30
Val Ile Cys Val Ala Asp Val Ser Ser Thr Asn Phe Asp Ser Leu Gln
35 40 45
Lys Ser Ile Ala Glu Ile Asn Gln Leu Thr Leu Val Gln Cys Thr Val
50 55 60
Leu Asp Val Thr Ser Ser Glu Asp Val Asn Gln Trp Leu Arg Asp Ile
65 70 75 80
Val Ser Ser His Gly Asn Leu His Gly Ala Ala Asn Ile Ala Gly Val
85 90 95
Ala Gln Gly Ala Gly Leu Arg Thr Thr Pro Thr Ile Leu Ala Glu Thr
100 105 110
Asp Ala Asp Trp Ser Arg Ile Leu Lys Val Asn Leu Asp Gly Val Phe
115 120 125
Tyr Cys Thr Arg Ala Gln Val Arg Ala Met Thr Asp Leu Pro Lys Gly
130 135 140
Asn Arg Ser Ile Val Asn Val Ala Ser Ile Ala Ala Phe Ser His Met
145 150 155 160
Pro Asp Val Tyr Ala Tyr Ser Thr Ser Lys Gln Ala Cys Val His Phe
165 170 175
Thr Ala Cys Val Ala Ala Asp Val Phe Pro Leu Gly Ile Arg Val Asn
180 185 190
Cys Val Ser Pro Gly Ile Thr Asn Thr Pro Leu Leu Pro Gln Phe Glu
195 200 205
Pro Asn Ala Lys Ser Leu Asp Glu Ile Gln Ala Leu Tyr Ala Ser Gln
210 215 220
Gly Leu Thr Met Cys Gly Ala Asp Glu Val Ala Arg Thr Ile Val Trp
225 230 235 240
Leu Leu Ser Glu Asp Ser His Pro Val Tyr Gly Ala Asn Ile Asn Val
245 250 255
Gly Ala Gly Val Pro
260
<210> 61
<211> 265
<212> PRT
<213> Trichophyton verrucosum
<400> 61
Met Ser Phe Val Ser Ser Lys Ile Phe Ala Ile Thr Gly Gly Ala Ser
1 5 10 15
Gly Ile Gly Ala Ala Thr Cys Arg Leu Leu Ala Lys Arg Gly Ala Ala
20 25 30
Thr Leu Cys Val Gly Asp Leu Cys Ser Glu Asn Met Lys Leu Leu Glu
35 40 45
Lys Asp Ile Lys Lys Ile Asn Pro Asp Thr Lys Val His Cys Thr Val
50 55 60
Leu Asp Val Ser Ser Ser Ser Asn Val Asp Glu Trp Ile Gln Asp Ile
65 70 75 80
Ile Thr Thr Phe Gly Asp Leu His Gly Ala Ala Asn Ile Ala Gly Ile
85 90 95
Ala Gln Gly Ala Gly Leu Arg Gln Ala Pro Thr Ile Leu Glu Asp Asp
100 105 110
Asp Gln Gln Trp Lys Lys Val Phe Gln Val Asn Leu Asp Gly Val Leu
115 120 125
Tyr Ser Thr Arg Ala Gln Val Arg Ala Met Lys Glu Phe Ser Ser Thr
130 135 140
Asn Pro Gly Asp Arg Ser Ile Val Asn Val Ala Ser Ile Ala Ser Met
145 150 155 160
Ser His Met Pro Asp Val Phe Ala Tyr Gly Thr Ser Lys Ala Gly Cys
165 170 175
Ala Tyr Phe Thr Thr Phe Glu Val Thr Leu Phe Ala Leu Gly Tyr Phe
180 185 190
Ser Asn Ile Met Gly Met Leu Glu Gly Ile Thr Arg Thr Pro Met Leu
195 200 205
Pro Arg Phe Val Pro Ser Ala Lys Thr Gln Glu Glu Val Glu Glu Thr
210 215 220
Tyr Lys Lys Glu Gly Phe Ser Val Ile Glu Ala Asp Asp Val Ala Arg
225 230 235 240
Thr Ile Val Trp Leu Leu Ser Glu Asp Ser Arg Pro Val Phe Gly Ala
245 250 255
Asn Ile Asn Val Gly Ala Cys Met Pro
260 265
<210> 62
<211> 261
<212> PRT
<213> Claviceps fusiformis
<400> 62
Met Ser Ser Val Ser Ala Lys Ile Phe Ala Val Thr Gly Gly Ala Ser
1 5 10 15
Gly Ile Gly Ala Ala Thr Cys Arg Leu Leu Ala Glu Arg Gly Ala Ala
20 25 30
Val Ile Cys Val Ala Asp Val Asn Cys Thr Asp Phe Ala Ser Leu Gln
35 40 45
Glu Ser Ile Ala Gln Ile Asn Ala Ser Thr Leu Val Gln Cys Thr Lys
50 55 60
Val Asp Val Ser Ser Val Gln Asp Val Asn Leu Trp Leu His Gly Ile
65 70 75 80
Val Ser Lys His Gly Asp Leu His Gly Ala Ala Asn Val Ala Gly Ile
85 90 95
Ala Gln Ala Ala Gly Leu Arg Thr Lys Leu Thr Ile Leu Glu Glu Asp
100 105 110
Asp Ala Glu Trp Arg Arg Val Leu Asp Ile Asn Leu Asn Gly Val Phe
115 120 125
Tyr Ser Thr Arg Ala Glu Val Lys Val Met Arg Asp Leu Pro Leu Ala
130 135 140
His Arg Ser Ile Val Asn Val Ala Ser Ile Ala Ala Phe Ser His Val
145 150 155 160
Pro Asp Val Tyr Ala Tyr Gly Thr Ser Lys Asn Ala Cys Ala Tyr Leu
165 170 175
Thr Thr Cys Ile Ala Ala Asp Val Phe Trp Ser Gly Ile Arg Val Asn
180 185 190
Cys Val Ser Pro Gly Ile Thr Asn Thr Pro Leu Leu Pro Gln Phe Glu
195 200 205
Pro Lys Val Lys Thr Leu Asp Glu Ile Arg Lys Leu Tyr Arg Glu Gln
210 215 220
Gly Tyr Pro Thr Ser Glu Ala Glu Asp Val Ala Arg Thr Ile Val Trp
225 230 235 240
Leu Leu Ser Glu Asp Ser Arg Pro Val Tyr Gly Ala Asn Ile Asn Val
245 250 255
Gly Ala Cys Pro Pro
260
<210> 63
<211> 246
<212> PRT
<213> Arthroderma benhamiae
<400> 63
Met Ala Ser Val Ser Ser Lys Ile Phe Ala Ile Thr Gly Gly Ala Ser
1 5 10 15
Gly Ile Gly Ala Ala Thr Cys Arg Leu Leu Ala Lys Arg Gly Ala Ala
20 25 30
Thr Leu Cys Val Gly Asp Leu Cys Ser Glu Asn Met Lys Gln Leu Glu
35 40 45
Asn Asp Ile Lys Glu Ile Asn Pro Asn Thr Lys Val His Cys Thr Val
50 55 60
Leu Asp Val Ser Ser Ser Ser Asn Val Asp Glu Trp Ile Lys Asp Ile
65 70 75 80
Ile Thr Thr Phe Gly Asp Leu His Gly Ala Ala Asn Ile Ala Gly Ile
85 90 95
Ala Gln Gly Ala Gly Leu Arg Gln Ala Pro Thr Ile Leu Glu Glu Asp
100 105 110
Asp Gln Gln Trp Lys Lys Val Phe Gln Val Asn Leu Asp Gly Val Leu
115 120 125
Tyr Ser Thr Arg Ala Gln Val Arg Ala Met Lys Glu Ser Ser Ser Thr
130 135 140
Asn Pro Gly Asp Arg Ser Ile Val Asn Val Ala Ser Ile Ala Ser Met
145 150 155 160
Ser His Met Pro Asp Val Phe Ala Tyr Gly Thr Ser Lys Ala Gly Cys
165 170 175
Ala Tyr Phe Thr Thr Cys Val Ser Gln Asp Val Met Pro Phe Gly Ile
180 185 190
Arg Ala Asn Thr Val Ser Pro Glu Glu Val Glu Glu Thr Tyr Lys Lys
195 200 205
Glu Gly Phe Ser Val Ile Glu Ala Asp Asp Val Ala Arg Thr Ile Val
210 215 220
Trp Leu Leu Ser Glu Asp Ser Arg Pro Val Phe Gly Ala Asn Ile Asn
225 230 235 240
Val Gly Ala Cys Met Pro
245
<210> 64
<211> 609
<212> PRT
<213> Paecilomyces divaricatus
<400> 64
Met Arg Tyr Val Leu Leu Ala Val Ala Gly Leu Leu Leu Gly Trp Leu
1 5 10 15
Ser Phe Thr Gly Lys Asp Ser Arg Pro Cys Arg Cys Arg Pro Trp Thr
20 25 30
Ser Cys Trp Pro Ser Glu Glu Thr Trp Arg Ala Phe Asn Lys Ser Ile
35 40 45
Asp Gly Asn Leu Ile Arg Leu Arg Pro Ile Ala His Val Cys His Glu
50 55 60
Pro Thr Phe Asn Gln Ser Ala Cys Asn Glu Leu Arg Arg Phe Ser Leu
65 70 75 80
Asp Ser Gly Trp Arg Ala Ala Gln Pro Gly Ala Leu Gln Asp Trp Val
85 90 95
Trp Glu Ser Gly Ser Asn Pro Asp Glu Ala Cys Leu Met Ala Ser Pro
100 105 110
Arg Glu Met Pro Cys His Gln Gly Arg Val Pro Leu Phe Ser Ala Ala
115 120 125
Val Asp Ser Ala Glu Gln Val Gln Arg Ala Val Arg Phe Ala Lys Arg
130 135 140
Tyr Asn Leu Arg Leu Val Ile Arg Asn Thr Gly His Asp Gly Ala Gly
145 150 155 160
Gln Ser Ser Gly Pro Asp Ser Phe Gln Ile His Thr His Arg Leu Gln
165 170 175
Ala Ile Gln Tyr His Thr Asp Phe Gln Pro Asn Gly Ser Leu Val Arg
180 185 190
Leu Gly Ser Ala Val Thr Val Gly Ala Gly Val Gln Leu Gly Asp Leu
195 200 205
Tyr Ala Arg Gly Ser Leu Glu Gly Tyr Thr Val Val Gly Gly Glu Cys
210 215 220
Pro Thr Val Gly Ala Ala Gly Gly Phe Leu Gln Gly Gly Gly Val Ser
225 230 235 240
Ser Phe Leu Ser His Ser Trp Gly Leu Ala Val Asp Asn Val Leu Glu
245 250 255
Phe Glu Met Val Thr Ala Gln Gly Asp Leu Val Val Ala Asn Ala His
260 265 270
Gln Asn Gln Asp Leu Phe Trp Ala Leu Arg Gly Gly Gly Gly Gly Thr
275 280 285
Phe Gly Val Val Thr Arg Ala Thr Leu Arg Ala Tyr His Asp Leu Pro
290 295 300
Val Val Val Ala Lys Leu Thr Ile Ala Ala Leu Asp Ala Asp Ala Ile
305 310 315 320
Phe Trp Thr Lys Gly Val Thr Thr Leu Leu Thr Ala Leu Gln Lys Phe
325 330 335
Asn Arg Glu Ser Val Ala Gly Gln Phe Leu Leu Lys Pro Gln Thr Glu
340 345 350
Ser Gly Ile Glu Ala Ser Ile Thr Met Tyr Phe Ile Asn Glu Thr Glu
355 360 365
Thr Ala Arg Val Asp Asp Arg Leu Asn Pro Leu Leu Thr Ile Leu Asn
370 375 380
Ser Asp Gly Ile Thr Ser Ala Arg Ser Ser Gln Phe Leu Pro Arg Leu
385 390 395 400
Ser Met Asp Leu Arg Thr Ala Pro Asp Ile Tyr Pro Glu Asn Tyr Gly
405 410 415
Ile Leu Met Ala Ser Val Leu Val Ser Asn Gln Leu Phe Asn Trp Ser
420 425 430
Glu Gly Pro Gln Arg Met Ala Glu Asn Phe Ala Arg Leu Pro Met Gly
435 440 445
Pro His Asp Leu Leu Phe Thr Ser Asn Leu Gly Gly Arg Val Ile Ala
450 455 460
Asn Gly Asp Val Val Asp Thr Ala Met His Pro Ala Trp Arg Ser Ala
465 470 475 480
Ala Gln Leu Ile Asn Leu Val Arg Ala Val Asp Pro Ser Ile Ser Gly
485 490 495
Lys Ser Thr Ala Leu Glu Gly Leu Thr Asn Ile Gln Met Pro Ile Leu
500 505 510
Tyr Ser Leu Asp Pro Ala Phe Arg Ala Ser Tyr Leu Asn Leu Ala Asp
515 520 525
Pro Asn Glu Lys Asp Ser Pro Gln Val Phe Trp Gly Glu Asn Tyr Gly
530 535 540
Arg Leu Leu Gln Ile Lys Arg Lys Trp Asp Ala Gly Asp Leu Phe Leu
545 550 555 560
Thr Lys Leu Gly Val Gly Ser Asp Asp Trp Glu Met Asp Gly Met Cys
565 570 575
Arg Lys Arg Gln Asn Leu Ile Asn Arg Ala Phe Lys Ser Val Pro Ser
580 585 590
Gln Val Gly Lys Thr Leu Trp Arg Leu Leu Glu Gly Ser Ser Asn Asp
595 600 605
Leu
<210> 65
<211> 612
<212> PRT
<213> Arthroderma otae
<400> 65
Met Asn Ser Asn Ser Gln Ile Arg Ser Ser Arg Leu Ile Glu Ala Leu
1 5 10 15
His Ala Tyr Tyr Ser Lys Tyr Leu Val Val Thr Ile Arg Pro Ile Arg
20 25 30
Tyr Leu Val Glu Pro Trp Gln Pro Cys Trp Pro Ser Glu Glu Leu Trp
35 40 45
Asn Ser Phe Asn Thr Ser Ile Asp Gly Lys Leu Gln Gln Leu Lys Pro
50 55 60
Ala Ala His Val Cys Tyr Glu Pro Asn Phe Asp Lys Gly Ala Cys Asp
65 70 75 80
Asp Leu Leu Arg Leu Ser Arg Asp Ser Gly Trp Arg Ala Ser His Pro
85 90 95
Gly Val Leu Gln Asp Trp Val Trp Glu Ala Gly Glu Ser Ala Asn Glu
100 105 110
Thr Cys Pro Met Gly Ser Leu Gln Thr Ala Thr Ala Ala Lys Ser Cys
115 120 125
His Gln Gly Arg Ile Pro Leu Tyr Ser Ala Thr Val Glu Ser Ala Gln
130 135 140
Gln Val Gln Gln Ala Val Arg Phe Ala Arg Arg His Asn Leu Arg Leu
145 150 155 160
Val Ile Arg Asn Thr Gly His Asp Leu Ala Gly Arg Ser Ser Ala Pro
165 170 175
Asp Ser Phe Gln Ile His Thr His Arg Leu Gln Glu Thr Gln Phe His
180 185 190
Thr Asp Leu Arg Leu Asn Gly Ser Thr Ala Ser Leu Gly Pro Ala Val
195 200 205
Thr Val Gly Ala Gly Val Met Met Gly Asn Leu Tyr Ala Arg Ala Ala
210 215 220
Arg Glu Gly Tyr Met Val Leu Gly Gly Asp Cys Pro Thr Val Gly Val
225 230 235 240
Ala Gly Gly Phe Leu Gln Gly Gly Gly Val Ser Asp Phe Leu Ser Leu
245 250 255
Asn Gln Gly Leu Gly Val Asp Asn Val Leu Glu Tyr Glu Ile Val Thr
260 265 270
Ala Asp Gly Glu Leu Leu Val Ala Asn Thr Leu Gln Asn Gln Glu Leu
275 280 285
Phe Trp Ala Leu Arg Gly Gly Gly Gly Gly Thr Phe Gly Val Val Thr
290 295 300
Arg Ala Thr Met Arg Val Phe Pro Asp Val Pro Ala Val Ile Ser Glu
305 310 315 320
Val Leu Leu Gln Ala Pro Gln Thr Asn Ser Ser Ser Trp Thr Glu Gly
325 330 335
Leu Ser Val Ile Leu Asn Ala Leu Gln Ser Leu Asn Arg Asp Asp Val
340 345 350
Gly Gly Gln Leu Val Ile Ala Val Gln Pro Glu Leu Ala Val Gln Ala
355 360 365
Ser Ile Lys Phe Phe Phe Leu Asn Ser Thr Glu Thr Thr Ile Ile Asp
370 375 380
Glu Arg Met Lys Ser Leu Leu Thr Asp Leu Asn Arg Ile Asp Ile Gln
385 390 395 400
Tyr Thr Leu Ser Ser Lys Ala Leu Pro His Phe Ser Ser Asn Tyr Arg
405 410 415
Gln Val Pro Asp Ile His Ser Asp Asn Asp Tyr Gly Val Ile Gly Ser
420 425 430
Thr Val Ala Ile Ser Lys Glu Leu Phe Asp Ser Ser Gln Gly Pro Gln
435 440 445
Lys Ile Ala Arg Ala Leu Ala Asn Leu Pro Met Ser Pro Gly Asp Leu
450 455 460
Leu Phe Thr Ser Asn Leu Gly Gly Arg Val Ile Ser Asn Gly Glu Ile
465 470 475 480
Ala Glu Thr Ser Met His Pro Ala Trp Arg Ala Ala Ser Gln Leu Leu
485 490 495
Asn Tyr Ile His Ala Val Gly Pro Ser Ile Glu Ser Arg Val Asn Ala
500 505 510
Leu Glu Arg Leu Thr Asn Val Gln Met Pro Met Leu Tyr Ala Ile Asp
515 520 525
Pro Asn Phe Arg Leu Ser Tyr Arg Asn Val Gly Asp Pro Asn Glu Lys
530 535 540
Asp Phe Gln Gln Val Tyr Trp Gly Ser Asn Tyr Lys Arg Leu Ser Gln
545 550 555 560
Ile Lys Lys Arg Trp Asp Ser Asp Gly Leu Phe Phe Ser Lys Leu Gly
565 570 575
Val Gly Ser Glu Leu Trp Asp Ser Glu Gly Met Cys Arg Lys Asn Gln
580 585 590
Ser Val Val Arg Gln Ala Val Asn Tyr Leu Met Ser Phe Ala Thr Ser
595 600 605
Met Val Glu Gly
610
<210> 66
<211> 607
<212> PRT
<213> Trichophyton tonsurans
<400> 66
Met His Ala Tyr Phe Leu Pro Ile Phe Gly Cys Cys Asn Ser Ala Asn
1 5 10 15
Ser Leu Pro Gly Gly Glu Thr Gln Pro Ala Ser Cys Arg Cys Arg Pro
20 25 30
Trp Glu Ser Cys Trp Pro Ser Glu Glu Leu Trp Asn Ser Phe Asn Thr
35 40 45
Ser Val Asp Gly Lys Leu His Arg Leu Arg Pro Ala Ala His Val Cys
50 55 60
Tyr Gly Pro Ser Phe Asn Lys Ser Ala Cys Asp Asn Ile Leu Leu Leu
65 70 75 80
Ser Arg Asp Ser Gly Trp Arg Ala Ser Asn Pro Gly Met Leu Gln Asp
85 90 95
Trp Val Trp Glu Ala Gly Glu Thr Ala Asn Glu Ser Cys Pro Val Gly
100 105 110
Ser Leu Arg Thr Ala Ser Ala Val Asn Ser Cys His Gln Gly Arg Ile
115 120 125
Pro Leu Phe Thr Val Gly Val Glu Ser Thr Lys Gln Val Gln Glu Ala
130 135 140
Val Arg Phe Ala Arg Lys His Asn Leu Arg Leu Val Ile Arg Asn Thr
145 150 155 160
Gly His Asp Leu Ala Gly Arg Ser Ser Ala Pro Asp Ser Phe Gln Ile
165 170 175
His Thr His Arg Leu Gln Glu Ile Arg Phe His Ala Asp Met Arg Leu
180 185 190
Asp Gly Ser Asn Thr Ser Leu Gly Pro Ala Val Thr Val Gly Ala Gly
195 200 205
Val Met Met Gly Asn Leu Tyr Ala Gln Ala Ala Arg His Gly Tyr Met
210 215 220
Val Leu Gly Gly Asp Cys Pro Thr Val Gly Val Val Gly Gly Phe Leu
225 230 235 240
Gln Gly Gly Gly Ile Ser Asp Phe Leu Ser Leu Asn Gln Gly Phe Gly
245 250 255
Val Asp Asn Val Leu Glu Tyr Glu Leu Val Thr Ala Asp Gly Glu Leu
260 265 270
Val Val Ala Asn Ala Leu Gln Asn Gln Asp Leu Phe Trp Ala Leu Arg
275 280 285
Gly Gly Gly Gly Gly Thr Phe Gly Val Val Thr Arg Ala Thr Met Arg
290 295 300
Ile Phe Pro Asp Ile Pro Val Val Ile Ser Glu Ile Leu Leu Glu Ala
305 310 315 320
Pro Gln Ala Ile Ser Ser Ser Trp Thr Glu Gly Leu Ser Ile Val Leu
325 330 335
Thr Ala Leu Gln Ser Leu Asn Arg Asp Asn Val Gly Gly Gln Leu Val
340 345 350
Ile Ala Val Leu Pro Lys Leu Ala Val Gln Ala Ser Ile Lys Phe Phe
355 360 365
Phe Leu Asp Val Thr Glu Ala Ala Ile Ile Asp Arg Arg Met Lys Pro
370 375 380
Phe Leu Thr Lys Leu Ser Arg Ala Asn Val Lys Tyr Thr Tyr Ser Ser
385 390 395 400
Lys Ser Leu Pro His Phe Ser Ser Asn Tyr Arg Gln Val Pro Asp Val
405 410 415
His Ser Asp Asn Asp Tyr Gly Val Leu Gly Ser Thr Val Ala Ile Ser
420 425 430
Gln Gln Leu Phe Asp Ser Pro Gln Gly Pro Glu Lys Val Ala Asn Ala
435 440 445
Leu Ala Asn Ile Pro Val Ser Val Gly Asp Leu Ile Phe Thr Ser Asn
450 455 460
Leu Gly Gly Arg Val Ile Arg Asn Gly Glu Leu Ala Glu Thr Ser Met
465 470 475 480
His Pro Ala Trp Arg Ser Ala Ser Gln Leu Ile Asn Tyr Val His Thr
485 490 495
Val Lys Pro Ser Ile Glu Gly Arg Ala Lys Ala Arg Glu Arg Leu Thr
500 505 510
Asn Thr Gln Met Pro Met Leu Tyr Ala Leu Asp Pro Asn Phe Lys Leu
515 520 525
Ser Tyr Arg Asn Val Gly Asp Pro Asn Glu Lys Asp Phe Gln Gln Ile
530 535 540
Tyr Trp Gly Pro Asn Tyr Gly Arg Leu Ser Asp Ile Lys Lys Lys Trp
545 550 555 560
Asp Thr Asp Asp Leu Phe Phe Ser Lys Leu Gly Val Gly Ser Glu Arg
565 570 575
Trp Asp Ser Glu Gly Met Cys Arg Lys Ser Arg Asp Ala Leu His Gln
580 585 590
Lys Ile Thr Arg Phe Ile Ser Phe Pro Thr Ser Ile Trp Arg Gly
595 600 605
<210> 67
<211> 605
<212> PRT
<213> Epichloe brachyelytri
<400> 67
Met Arg Tyr Leu Val Thr Phe Ala Val Gly Leu Leu Leu Ser Trp Gly
1 5 10 15
Phe Leu Ser Leu Leu Gln Gly Arg Pro Ser Cys Arg Cys Arg Pro Trp
20 25 30
Glu Ala Cys Trp Pro Asn Ser His Gln Trp Thr Ser Leu Asn Ala Ser
35 40 45
Ile Asp Gly Asn Leu Val His Val Gln Pro Val Gly Ser Val Cys His
50 55 60
Asp Pro Glu Tyr Asp Ala Val Ala Cys Gly Asp Val Leu Asp Leu Ser
65 70 75 80
Arg Asn Ser Gly Trp Arg Ala Ser Asn Pro Ala Thr Leu Gln Asp Trp
85 90 95
Ile Trp Glu Thr Gly Ser Gly Asp Asn Glu Ser Cys Leu Leu Val Ser
100 105 110
Ser Ser Glu Arg Ser Gln Glu Ile Pro Cys His Gln Gly Arg Leu Pro
115 120 125
Leu Tyr Ser Ala Ala Val Lys Ser Thr Ala His Val Gln Ser Val Val
130 135 140
Arg Phe Ala Lys Asp His Asn Leu Arg Leu Val Ile Lys Asn Thr Gly
145 150 155 160
His Asp Ala Thr Gly Arg Ser Ala Ala Pro Asp Ser Leu Gln Ile His
165 170 175
Thr Tyr Phe Leu Lys Asp Ile His Tyr His Asp Asn Phe Phe Ala Gln
180 185 190
Gly Asp Ala Thr Gly Ser Gly Pro Ala Val Thr Leu Gly Ala Gly Val
195 200 205
Val His Ser Glu Val Tyr Lys His Gly Ile Asp His Lys Tyr Ser Val
210 215 220
Val Gly Gly Glu Cys Pro Thr Val Gly Ile Val Gly Gly Phe Leu Gln
225 230 235 240
Gly Gly Gly Val Ser Ser Trp Ser Gly Phe Thr Arg Gly Leu Ala Val
245 250 255
Asp Asn Val Leu Glu Tyr Gln Val Val Thr Ala Asn Ala Glu Leu Val
260 265 270
Ile Ala Asn Glu His Gln Asn Gln Asp Leu Phe Trp Ala Leu Arg Gly
275 280 285
Gly Gly Gly Gly Thr Phe Gly Val Val Thr Gln Ala Thr Val Arg Ala
290 295 300
Phe Pro Asp Asp Pro Thr Val Val Ser Thr Leu Val Leu Thr Ser Thr
305 310 315 320
Arg Ala Asp Thr Ser Phe Trp Glu Lys Ala Ile Ser Arg Leu Leu Ser
325 330 335
Ile Leu Arg Ser Cys Asn Gln Gln Asn Val His Gly Gln Leu Ile Ile
340 345 350
Thr Arg Pro Ser Glu Asp Ile Leu Asn Ala Gly Leu Thr Leu His Phe
355 360 365
Ser Asn Met Thr Asn Val Ser His Ala Glu Thr Leu Leu Gln Pro His
370 375 380
Ile Ala Ser Leu Arg Glu Glu Gln Ile Ser Ala Ala Leu Ser Cys Lys
385 390 395 400
Phe Val Ala Asn Ile Asn Ser Glu Leu Arg Leu Asp Ala Asp Ile His
405 410 415
Pro Arg Gly Ile Gly Thr Leu Gln Thr Ser Met Met Ile Ser Asp Glu
420 425 430
Leu Phe Gly Ser Ser Glu Gly Pro Ser Thr Val Ala Gln Val Phe Gly
435 440 445
Lys Leu Pro Ile Gly Gln Asn Asp Leu Leu Phe Thr Ser Asn Leu Gly
450 455 460
Gly Arg Ile Ala Ala Asn Lys Ala Leu Asp Thr Ala Ile His Pro Ala
465 470 475 480
Trp Arg Ser Ser Ala His Leu Val Thr Tyr Val Arg Ser Val Glu Pro
485 490 495
Ser Ile Glu Ala Lys Lys Leu Ala Leu Asp Glu Ile Thr Asn Lys Tyr
500 505 510
Met Pro Ile Leu Tyr Ser Met Gln Pro Ser Phe Lys Val Ser Tyr Arg
515 520 525
Asn Leu Gly Asp Pro Asn Glu Lys Asn Tyr Gln Glu Val Phe Trp Gly
530 535 540
Glu Lys Ala Tyr Lys Arg Leu Ala Ser Ile Lys Ala Lys Leu Asp Pro
545 550 555 560
Asp Gly Leu Phe Ile Ser Lys Leu Gly Val Gly Ser Glu Asp Trp Asp
565 570 575
Ala Glu Gly Met Cys Tyr Gln Pro Gln Asn Arg Val Ser Gln Pro Leu
580 585 590
Arg Ser Leu Gln Tyr Phe Leu Ser Val Leu Gln Asp Thr
595 600 605
<210> 68
<211> 604
<212> PRT
<213> Trichophyton verrucosum
<400> 68
Met Gln Phe Leu Leu Trp Ser Thr Gly Leu Val Ala Leu Leu Ser Trp
1 5 10 15
Leu Ile Tyr Thr Gln Glu Thr Gln Ser Ala Ser Cys Arg Cys Arg Pro
20 25 30
Trp Glu Ser Cys Trp Pro Ser Glu Glu Leu Trp Asn Ser Phe Asn Thr
35 40 45
Ser Val Asp Gly Lys Leu His Arg Leu Arg Pro Ala Ala His Val Cys
50 55 60
Tyr Gly Pro Ser Phe Asn Arg Ser Ala Cys Asp Asn Ile Leu Leu Leu
65 70 75 80
Ser Arg Asp Ser Gly Trp Arg Ala Ser Asn Pro Gly Val Leu Gln Asp
85 90 95
Trp Val Trp Glu Ala Gly Glu Thr Ala Asn Glu Ser Cys Pro Val Gly
100 105 110
Ser Leu Arg Thr Ala Ser Ala Val Asn Ser Cys His Gln Gly Arg Ile
115 120 125
Pro Leu Phe Thr Val Gly Val Glu Ser Thr Lys Gln Val Gln Glu Ala
130 135 140
Val Arg Phe Ala Arg Lys His Lys Leu Arg Leu Val Ile Arg Asn Thr
145 150 155 160
Gly His Asp Leu Ala Gly Arg Ser Ser Ala Pro Asp Ser Phe Gln Ile
165 170 175
His Thr His Arg Leu Gln Glu Ile Gln Phe His Ala Asp Met Arg Leu
180 185 190
Asp Gly Ser Asn Thr Ser Leu Gly Pro Ala Val Thr Val Gly Ala Gly
195 200 205
Val Met Met Gly Asp Leu Tyr Ala Gln Ala Ala Arg His Gly Tyr Met
210 215 220
Val Leu Gly Gly Asp Cys Pro Thr Val Gly Val Val Gly Gly Phe Leu
225 230 235 240
Gln Gly Gly Gly Ile Ser Asp Phe Leu Ser Leu Asn Gln Gly Phe Gly
245 250 255
Val Asp Asn Val Leu Glu Tyr Glu Val Val Thr Ala Asp Gly Glu Leu
260 265 270
Val Val Ala Asn Ala Leu Gln Asn Gln Asp Leu Phe Trp Ala Leu Arg
275 280 285
Gly Gly Gly Gly Gly Thr Phe Gly Val Val Thr Arg Ala Thr Met Arg
290 295 300
Val Phe Pro Asp Val Pro Val Val Ile Ser Glu Ile Leu Leu Glu Ala
305 310 315 320
Pro Gln Ala Ile Ser Ser Ser Trp Thr Gln Gly Leu Ser Ile Val Leu
325 330 335
Thr Ala Leu Gln Ser Leu Asn His Asp Asn Val Gly Gly Gln Leu Val
340 345 350
Ile Ala Val Leu Pro Asn Leu Ala Val Gln Ala Ser Ile Lys Phe Phe
355 360 365
Phe Leu Asp Ala Thr Glu Ala Ala Val Ile Asp Arg Arg Met Lys Pro
370 375 380
Phe Leu Thr Lys Leu Ser Arg Ala Asn Val Lys Tyr Thr Tyr Ser Ser
385 390 395 400
Lys Asn Leu Pro His Phe Ser Ser Asn Tyr Arg Gln Val Pro Asp Ile
405 410 415
His Ser Asp Asn Asp Tyr Gly Val Leu Gly Ser Thr Val Ala Ile Ser
420 425 430
Gln Gln Leu Phe Asp Ser Pro Gln Gly Pro Glu Lys Val Ala Lys Ala
435 440 445
Leu Ala Asn Leu Pro Val Ser Ala Gly Asp Leu Ile Phe Thr Ser Asn
450 455 460
Leu Gly Gly Arg Val Ile Arg Asn Gly Glu Leu Ala Glu Thr Ser Met
465 470 475 480
His Pro Ala Trp Arg Ser Ala Ser Gln Leu Ile Asn Tyr Val His Thr
485 490 495
Val Glu Pro Ser Ile Glu Gly Arg Ala Lys Ala Arg Glu Arg Leu Thr
500 505 510
Asn Thr Gln Met Pro Met Leu Tyr Ala Leu Asp Pro Asn Ile Lys Leu
515 520 525
Ser Tyr Arg Asn Val Gly Asp Pro Asn Glu Lys Asp Phe Gln Gln Ile
530 535 540
Tyr Trp Gly Pro Asn Tyr Gly Arg Leu Ser Asn Ile Lys Lys Lys Trp
545 550 555 560
Asp Thr Asp Asp Leu Phe Phe Ser Lys Leu Gly Val Gly Ser Glu Arg
565 570 575
Trp Asp Ser Glu Glu Ser Asp Glu Val Tyr Phe Phe Ser Tyr Phe Tyr
580 585 590
Leu Glu Gly Ile Asn Thr Leu Lys Val Ala Arg Arg
595 600
<210> 69
<211> 605
<212> PRT
<213> Neotyphodium lolii
<400> 69
Met Arg His Phe Val Thr Phe Val Val Gly Phe Leu Leu Ser Trp Gly
1 5 10 15
Phe Leu Ser Ser Leu Gln Asp Pro Pro Ser Cys Arg Cys Arg Pro Trp
20 25 30
Glu Pro Cys Trp Pro Asp Ser His Gln Trp Thr Ser Leu Asn Ala Ser
35 40 45
Ile Asp Gly Asn Leu Val His Val Gln Pro Val Gly Ser Val Cys His
50 55 60
Asp Pro His Tyr Asp Ala Val Ala Cys Gly Asp Val Leu Asp Leu Ser
65 70 75 80
Arg Asn Ser Gly Trp Arg Ala Ser Asn Pro Ala Thr Leu Gln Asp Trp
85 90 95
Ile Trp Glu Thr Gly Ser Gly Asp Asn Glu Ser Cys Leu Leu Val Ser
100 105 110
Ser Ser Glu Arg Pro Gln Glu Ile Pro Cys His Gln Gly Arg Leu Pro
115 120 125
Leu Tyr Ser Ala Ala Val Lys Ser Thr Ala His Val Gln Gly Val Ile
130 135 140
Arg Phe Ala Lys Asp His Asn Leu Arg Leu Val Ile Lys Asn Thr Gly
145 150 155 160
His Asp Ala Thr Gly Arg Ser Ala Ala Pro Asp Ser Leu Gln Ile His
165 170 175
Thr Tyr Phe Leu Lys Asp Ile His Tyr Asp Asp Asn Phe Leu Val His
180 185 190
Gly Asp Ala Thr Gly Ser Gly Pro Ala Val Thr Leu Gly Ala Gly Val
195 200 205
Val His Ser Glu Val Tyr Lys His Gly Ile Asp His Lys Tyr Ser Val
210 215 220
Val Gly Gly Glu Cys Pro Thr Val Gly Ile Val Gly Gly Phe Leu Gln
225 230 235 240
Gly Gly Gly Val Ser Ser Trp Ser Gly Phe Thr Arg Gly Leu Ala Val
245 250 255
Asp Asn Val Leu Glu Tyr Gln Val Val Thr Ala Asn Ala Glu Leu Val
260 265 270
Ile Ala Asn Glu His Gln Asn Gln Asp Leu Phe Trp Ala Leu Arg Gly
275 280 285
Gly Gly Gly Gly Thr Phe Gly Val Val Thr Gln Ala Thr Val Arg Ala
290 295 300
Phe Pro Asp Asp Pro Thr Val Val Ser Thr Leu Val Leu Ser Ser Thr
305 310 315 320
Arg Ala Asp Thr Ser Phe Trp Val Lys Ala Ile Ser Arg Leu Leu Ser
325 330 335
Ile Leu Arg Ser Cys Asn Gln Gln Asn Val His Gly Gln Leu Ile Ile
340 345 350
Thr Arg Pro Ser Val Asp Ile Leu Asn Ala Gly Leu Thr Leu His Phe
355 360 365
Ser Asn Met Thr Asn Val Leu His Ala Glu Thr Leu Leu Gln Pro His
370 375 380
Ile Ala Ser Leu Ser Glu Asp Gln Ile Ser Thr Thr Leu Thr Ser Lys
385 390 395 400
Phe Val Ala Asn Ile Asn Ser Glu Leu Arg Leu Asp Ala Asp Ile His
405 410 415
Pro Arg Gly Ile Gly Thr Leu Gln Thr Ser Met Met Ile Ser Asn Glu
420 425 430
Leu Phe Gly Ser Ser Glu Gly Pro Leu Thr Val Ala Gln Val Phe Gly
435 440 445
Lys Leu Pro Ile Gly Pro Asn Asp Leu Leu Phe Thr Ser Asn Leu Gly
450 455 460
Gly Cys Ile Ala Ala Asn Lys Gly Leu Asp Thr Ala Ile His Pro Ala
465 470 475 480
Trp Arg Ser Ser Ala His Leu Val Thr Tyr Val Arg Ser Val Glu Pro
485 490 495
Ser Ile Glu Ala Lys Lys Leu Ala Leu Lys Glu Ile Thr Asn Lys Tyr
500 505 510
Met Pro Ile Leu Tyr Ser Met Gln Pro Ser Phe Lys Val Ser Tyr Arg
515 520 525
Asn Leu Gly Asp Pro Asn Glu Lys Asn Tyr Gln Glu Val Phe Trp Gly
530 535 540
Glu Lys Val Tyr Lys Arg Leu Ala Ser Ile Lys Ala Lys Leu Asp Pro
545 550 555 560
Asp Gly Leu Phe Ile Ser Lys Leu Gly Val Gly Ser Glu Asp Trp Asp
565 570 575
Thr Glu Gly Met Cys Tyr Gln Pro Gln Asn Arg Val Ser Gln Pro Leu
580 585 590
Arg Ser Leu Lys Tyr Phe Leu Ser Val Leu Lys Asp Thr
595 600 605
<210> 70
<211> 628
<212> PRT
<213> Aspergillus fumigatus
<400> 70
Met Ser His Arg Ile Leu Cys Val Ala Phe Cys Val Cys Ser Leu Val
1 5 10 15
Ala Val Ser Ser Ile Tyr Ser Pro Pro Phe Asn His Pro Ile Val Tyr
20 25 30
Ser Asn Asn Ala Ile Cys Phe Gln Leu Ser Thr Trp Arg Ile Pro Gln
35 40 45
Leu Tyr Leu Ala Val Leu Ile Arg Arg Phe Asp Leu Ser Leu Ser Thr
50 55 60
Arg Gly Val Met Leu Ala Val Thr Val Gly Val Gly Ser Ser Gln His
65 70 75 80
His Ser Gln Arg Ser Gly Leu Ser Leu Thr Phe Ala Met Thr Pro Ser
85 90 95
Met Ile Thr Leu Leu Val Lys Thr Cys Val Ile Ser Gln Arg Thr Ala
100 105 110
Val Gly Glu Pro His Arg Pro Gly Trp Val Trp Glu Val Gly Arg Thr
115 120 125
Glu Asp Glu Thr Cys His Ala His Ser Pro Arg Gly Ala Pro Cys His
130 135 140
Gln Gly Arg Ile Pro Leu Tyr Ser Ala Ala Val Glu Ser Val Asp Gln
145 150 155 160
Ile Gln Val Ala Val Arg Phe Ala Gln Arg His Arg Leu Arg Leu Val
165 170 175
Val Arg Asn Thr Gly His Asp Thr Ala Gly Arg Ser Ser Gly Ser Asp
180 185 190
Ser Phe Gln Ile His Cys His Arg Met Lys Gln Ile Glu Tyr His Asp
195 200 205
Asn Phe Arg Ala Leu Gly Ser Asp Ile Asp Arg Gly Pro Ala Val Ser
210 215 220
Val Gly Ala Gly Val Thr Leu Gly Glu Met Tyr Ala Arg Gly Ala Arg
225 230 235 240
Asp Gly Trp Val Val Val Gly Gly Glu Cys Pro Thr Val Gly Ala Ala
245 250 255
Gly Gly Phe Leu Gln Gly Gly Gly Val Ser Ser Phe His Ser Phe Ile
260 265 270
Asp Gly Leu Ala Val Asp Asn Val Leu Glu Phe Glu Val Val Thr Ala
275 280 285
Lys Gly Asp Val Val Val Ala Asn Asp His Gln Asn Pro Asp Ile Phe
290 295 300
Trp Ala Leu Arg Gly Gly Gly Gly Gly Thr Phe Gly Ile Val Thr Arg
305 310 315 320
Ala Thr Met Arg Val His Leu Asn Ser Pro Val Cys Val Ser Glu Val
325 330 335
Ala Val Ser Gly Leu Arg Asn Asn Ser Leu Leu Trp Thr Lys Gly Ile
340 345 350
Thr Gly Leu Phe Ser Ile Leu Arg Ser Phe Asn Gln Gln Gly Ile Pro
355 360 365
Gly Gln Phe Ile Leu Arg Pro Leu Ser Lys Asp Gln Val Asn Ala Ser
370 375 380
Leu Thr Leu Tyr Ser Leu Asn Thr Asp Asp Thr Arg Arg Ser Ala Glu
385 390 395 400
Asn Met Leu Ser Ile Arg Asn Ile Leu Glu Ser Thr Thr Leu Pro Phe
405 410 415
Thr Leu Ala Ser Arg Cys Leu Pro Lys Ile Ser Asp Ala Leu Arg Lys
420 425 430
Gly Pro Asp Met Leu Pro Val Asn Tyr Gly Ile Ile Thr Gly Ser Val
435 440 445
Leu Val Ser Glu Asp Leu Phe Asn Ser Glu Glu Gly Pro Leu His Leu
450 455 460
Ala Lys Gln Leu Glu His Phe Pro Met Gly Pro Met Asp Leu Leu Phe
465 470 475 480
Thr Ser Asn Leu Gly Gly Asn Val Ser Ala Asn Thr Gly Lys Lys His
485 490 495
Arg Asp Thr Ser Met His Pro Gly Trp Arg Gln Ala Ala His Leu Ile
500 505 510
Asn Phe Val Arg Ser Val Ser Thr Pro Thr Ala His Glu Lys Ala Arg
515 520 525
Ser Leu Glu Glu Leu His Ser Val Gln Met Arg Gln Leu Tyr Asp Ile
530 535 540
Glu Pro Asp Phe Arg Val Ser Tyr Arg Asn Leu Gly Asp Pro Leu Glu
545 550 555 560
Ser Asp Ala Ala Gln Val Tyr Trp Gly Pro Asn Tyr Lys Arg Leu Leu
565 570 575
Glu Ile Lys Arg Lys Trp Asp Pro Glu Asp Leu Phe Phe Ser Gln Leu
580 585 590
Gly Val Gly Ser Glu Gly Trp Thr Glu Asp Gln Met Cys Lys Arg Gln
595 600 605
Gln Arg Leu Gln Gln Met Leu Gln Tyr Leu Met Ser Ser Ile Ala Gln
610 615 620
Arg Val Tyr Arg
625
<210> 71
<211> 600
<212> PRT
<213> Periglandula ipomoeae
<400> 71
Met His Arg Leu Leu Gly Pro Val Ala Cys Ile Thr Leu Ala Gly Ala
1 5 10 15
Val Phe Trp Ala Gln Ser Gly His Cys Arg Cys Arg Pro Trp Glu Pro
20 25 30
Cys Trp Pro Ser Thr Ala Gln Trp Glu Ala Phe Asn Glu Ser Ile Gln
35 40 45
Gly Asn Leu Val Arg Ile Arg Pro Ile Ala Ser Val Cys His Gly Leu
50 55 60
Glu Tyr Asp Ala Ile Ala Cys Ala Asn Leu Ser Asn Leu Ile Val Asp
65 70 75 80
Ser Gly Trp Arg Ala Ser Asn Pro Ser Thr Leu Gln Asp Trp Val Trp
85 90 95
Glu Ser Gly Asn Val Ala Glu Glu Ala Cys His Ile Met Thr Ser Pro
100 105 110
Gly Glu Gln Arg Gly Ser Ala Cys His Gln Gly Arg Leu Pro Leu Tyr
115 120 125
Ser Ala Val Val Gln Ser Thr Ser Asp Ile Gln Ser Ser Val Arg Phe
130 135 140
Ala Ser Asp His Asn Leu Arg Leu Val Val Lys Asn Thr Gly His Asp
145 150 155 160
Cys Ala Gly Arg Ser Ser Ser Pro Asn Ser Phe Gln Ile His Thr Asn
165 170 175
Leu Leu Lys Ser Ile Ser Phe His Glu Asn Phe Val Ala Arg Gly Ser
180 185 190
Thr Thr Cys Ser Gly Pro Ala Val Thr Leu Gly Ala Gly Val Met His
195 200 205
Trp Glu Val Tyr Ala His Gly Val Glu Asn Gly Tyr Thr Ile Val Gly
210 215 220
Gly Glu Cys Pro Thr Val Gly Ala Val Gly Gly Phe Leu Gln Gly Gly
225 230 235 240
Gly Val Ser Ser Ile His Ser Phe Thr Lys Gly Leu Ala Val Asp Asn
245 250 255
Val Leu Glu Tyr Gln Val Val Thr Pro Asn Ala Asp Leu Val Thr Ala
260 265 270
Asn Glu His Glu Asn Gln Asp Leu Tyr Trp Ala Leu Lys Gly Gly Gly
275 280 285
Gly Gly Thr Phe Gly Ile Val Thr Gln Ala Thr Val Arg Val Tyr Pro
290 295 300
Asp Asp Pro Val Val Val Thr Ala Thr Met Ile Lys Thr Ala Phe Ala
305 310 315 320
Asn Thr Leu Phe Trp Thr Glu Gly Val Arg Glu Leu Leu Arg Leu Leu
325 330 335
Gln Tyr Phe Asn Gln Leu His Ile Pro Gly Gln Leu Val Val Thr Gly
340 345 350
Pro Glu Lys Asp Ser Ile Gln Ala Thr Leu Glu Leu His Phe Ala Asn
355 360 365
Met Thr Asp Glu Ala His Ala Met Lys Leu Leu Ser Ser Glu Leu Arg
370 375 380
Pro Leu Ser Ile His His Ile Ser Thr Ser Thr Ser Val Arg Val Gln
385 390 395 400
Lys Arg Ala Ser Ser Glu Leu Arg Met Lys Pro Asp Ile Tyr Pro Pro
405 410 415
Gln Tyr Gly Ile Leu Gln Gly Ser Val Leu Ile Ser Thr Ala Val Phe
420 425 430
Gln Ser Ala Glu Gly Pro Thr Leu Ile Ala Arg Lys Phe Ser Glu Leu
435 440 445
Thr Val Arg Ser Asn Asp Ile Leu Phe Thr Ser Asn Leu Gly Gly Arg
450 455 460
Val Ser Lys Asn Ile Ala Ile Asp Val Pro Leu His Pro Ala Trp Arg
465 470 475 480
Glu Ala Ala Gln Leu Val Thr Phe Val Arg Ala Val Gly Pro Ser Ile
485 490 495
Glu Gly Lys Leu Ser Ala Leu Glu Asn Leu Thr Ser Gln Asp Ile Pro
500 505 510
Ile Leu Tyr Ser Val Asp Pro Asn Ala Lys Ile Ser Tyr Arg Asn Leu
515 520 525
Gly Asp Pro Gln Glu Gln Glu Phe Gln Ala Arg Tyr Trp Gly Leu Asp
530 535 540
Asn Tyr Ala Arg Leu Ala Ala Ile Lys Ala Lys Trp Asp Pro Asn Gly
545 550 555 560
Leu Phe Ile Thr Arg Ser Gly Val Gly Ser Glu Asp Trp Asp Val Glu
565 570 575
Gly Ile Cys Arg Lys Ser Arg Gly Leu Ile Ala Gln Ala Leu Glu Leu
580 585 590
Leu Lys Leu Gly Pro Tyr Ile Tyr
595 600
<210> 72
<211> 531
<212> PRT
<213> Arthroderma gypseum
<400> 72
Met Leu Leu Leu Ser Arg Asn Ser Gly Trp Arg Ala Ser Asn Pro Gly
1 5 10 15
Val Leu Gln Asp Trp Val Trp Glu Ala Gly Glu Thr Ala Asn Glu Thr
20 25 30
Cys Pro Val Gly Ser Leu Arg Thr Val Ser Ala Val Asn Ser Cys His
35 40 45
Gln Gly Arg Ile Pro Leu Tyr Ser Val Gly Val Glu Ser Ala Gln Gln
50 55 60
Val Gln Lys Ala Val Arg Phe Ala Arg Arg His Asn Leu Arg Leu Val
65 70 75 80
Ile Arg Asn Thr Gly His Asp Leu Ala Gly Arg Ser Ser Ala Pro Asp
85 90 95
Ser Phe Gln Ile His Thr His Arg Leu Gln Glu Ile His Phe His Thr
100 105 110
Asn Leu Arg Leu Asn Gly Ser Asn Ala Ser Leu Gly Pro Ala Val Thr
115 120 125
Val Gly Ala Gly Val Met Met Gly Asn Leu Tyr Ser Gln Ala Ala Arg
130 135 140
Asn Gly Tyr Met Val Leu Gly Gly Asp Cys Pro Thr Val Gly Val Val
145 150 155 160
Gly Gly Phe Leu Gln Gly Gly Gly Val Ser Asp Phe Leu Ser Leu Asn
165 170 175
Gln Gly Leu Gly Val Asp Asn Val Leu Glu Tyr Glu Val Val Thr Ala
180 185 190
Asp Gly Glu Ile Val Val Ala Asn Ala Leu Gln Asn Gln Asp Leu Phe
195 200 205
Trp Ala Leu Arg Gly Gly Gly Gly Gly Thr Phe Gly Val Val Thr Arg
210 215 220
Ala Thr Met Arg Val Phe Pro Asp Val Pro Val Val Ile Ser Glu Ile
225 230 235 240
Leu Leu Glu Ala Ser Lys Ser Asn Ser Ser Ser Trp Thr Gln Gly Leu
245 250 255
Ser Val Val Leu Thr Ala Leu Gln Ser Leu Asn Arg Glu Asn Val Gly
260 265 270
Gly Gln Leu Val Ile Val Val Leu Pro Lys Leu Ala Val Gln Ala Ser
275 280 285
Ile Lys Phe Phe Phe Leu Asp Ala Thr Glu Thr Ala Ala Ile Asp His
290 295 300
Arg Met Lys Ser Phe Leu Thr Glu Leu Ser Arg Ala Asn Val Lys Tyr
305 310 315 320
Ala Tyr Ser Ser Lys Gly Leu Pro His Phe Ser Ser Asn Tyr Arg Gln
325 330 335
Val Pro Asp Ile His Ala Asp Asn Asp Tyr Gly Val Leu Gly Ser Thr
340 345 350
Val Ala Ile Ser Gln Gln Leu Phe Asp Ser Pro Gln Gly Pro Glu Lys
355 360 365
Val Ala Asn Ala Leu Ala Asn Leu Pro Met Ser Pro Gly Asp Leu Leu
370 375 380
Phe Thr Ser Asn Leu Gly Gly Arg Val Ile Leu Asn Gly Gly Leu Ala
385 390 395 400
Glu Thr Ser Met His Pro Ala Trp Arg Ala Ala Ser Gln Leu Leu Asn
405 410 415
Tyr Val His Thr Val Glu Pro Ser Ile Glu Gly Arg Ala Lys Ala Arg
420 425 430
Glu Lys Leu Thr Asn Ile Gln Met Pro Met Leu Tyr Ala Ile Asp Pro
435 440 445
Lys Phe Lys Leu Ser Tyr Arg Asn Val Gly Asp Pro Asn Glu Lys Asn
450 455 460
Phe Gln Gln Val Tyr Trp Gly Ser Asn Tyr Gly Arg Leu Ser Asp Ile
465 470 475 480
Lys Lys Lys Trp Asp Thr Thr Asp Leu Phe Phe Ser Lys Leu Gly Ile
485 490 495
Gly Ser Glu Lys Trp Asp Ser Glu Gly Met Cys Arg Lys His Gln Glu
500 505 510
Ala Phe Tyr Gln Lys Met Arg Arg Phe Ile Ser Leu Thr Thr Phe Thr
515 520 525
Leu Lys Asn
530
<210> 73
<211> 595
<212> PRT
<213> Claviceps purpurea
<400> 73
Met Tyr Tyr Leu Leu Gly Pro Val Ala Cys Phe Ala Leu Ile Ala Ser
1 5 10 15
Val Leu Trp Ala Pro Ser Gly Arg Cys Arg Cys Arg Pro Trp Glu Pro
20 25 30
Cys Trp Pro Ser Ala Ala Asp Trp His Val Leu Asn Asp Ser Leu Gln
35 40 45
Gly Ser Leu Val Arg Ile Arg Pro Val Ala Ser Val Cys His Gly Ser
50 55 60
Glu Tyr Asp Val Ala Ala Cys Ala Asn Leu Ser Ser Leu Val Val Asp
65 70 75 80
Ser Gly Trp Arg Ala Ser Asn Pro Asn Thr Leu Gln Asp Trp Val Trp
85 90 95
Glu Ile Gly Asn Gly Val Glu Glu Phe Cys Pro Tyr Thr Ile Ser Pro
100 105 110
Met Glu Arg Arg Gln Ser Ile Cys Pro Gln Gly Arg Leu Pro Phe Tyr
115 120 125
Ser Ala Val Val Arg Ser Thr Ser Asp Ile Gln Ala Ser Val Arg Phe
130 135 140
Ala Ser Arg His Asn Leu Arg Leu Val Ile Lys Asn Thr Gly His Asp
145 150 155 160
Ser Ala Gly Arg Ser Ser Ala Pro His Ser Phe Gln Ile His Thr Ser
165 170 175
Leu Leu Gln Asn Ile Ser Leu His Lys Asn Phe Ile Ala Arg Gly Ser
180 185 190
Thr Thr Gly Arg Gly Pro Ala Val Thr Leu Gly Ala Gly Val Met Gln
195 200 205
Trp Gln Ala Tyr Val His Gly Ala Lys Asn Gly Tyr Thr Ile Leu Gly
210 215 220
Gly Glu Cys Pro Thr Val Gly Ala Ile Gly Gly Phe Leu Gln Gly Gly
225 230 235 240
Gly Val Ser Ser Ile His Ser Phe Thr Arg Gly Leu Ala Val Asp Gln
245 250 255
Val Leu Glu Tyr Gln Val Val Ser Ala Lys Gly Asp Leu Ile Thr Ala
260 265 270
Asn Glu Asp Asn Asn Gln Asp Leu Phe Trp Ala Leu Lys Gly Gly Gly
275 280 285
Gly Gly Thr Phe Gly Val Val Thr Glu Ala Thr Val Arg Val Phe Ser
290 295 300
Asp Asp Pro Val Thr Val Thr Ser Thr Lys Ile Glu Ala Ala Ala Ala
305 310 315 320
Asn Val Leu Phe Trp Lys Glu Gly Val His Glu Leu Leu Arg Leu Leu
325 330 335
Gln Arg Phe Asn Asn Leu His Val Ala Gly Gln Leu Val Ile Ser Ala
340 345 350
Pro Thr Lys Asp Ser Leu Gln Ala Gly Leu Glu Leu His Phe Ala Asn
355 360 365
Leu Thr Asp Glu Thr Gln Ala Ile Gln Leu Leu Arg Ser Glu Ala Arg
370 375 380
Ala Leu Glu Thr His Gly Ile Ser Ala Ser Thr Ser Val Arg Val Gln
385 390 395 400
Arg Lys Ala Ser Ser Glu Leu Arg Met Lys Pro Asp Leu Tyr Pro Pro
405 410 415
His Tyr Gly Ile Leu Glu Ala Ser Val Leu Ile Ser Ala Ala Thr Phe
420 425 430
His Ala Asn Asp Gly Pro Ala Leu Ile Ala Ser Lys Leu Ser Gly Leu
435 440 445
Thr Leu Lys Pro Asn Asp Ile Leu Phe Thr Ser Asn Leu Gly Gly Arg
450 455 460
Val Ser Glu Asn Thr Ala Ile Glu Ile Ala Leu His Pro Ala Trp Arg
465 470 475 480
Glu Ala Ala Gln Leu Val Thr Leu Val Arg Val Val Glu Pro Ser Ile
485 490 495
Glu Gly Lys Leu Ser Ala Leu Asn Asp Leu Thr Ala Arg Asp Val Pro
500 505 510
Ile Leu Tyr Ser Ile Asp Pro Ala Ala Lys Ile Ser Tyr Arg Asn Leu
515 520 525
Gly Asp Pro Gln Glu Lys Glu Phe Gln Ala Arg Tyr Trp Gly Ala Asp
530 535 540
Asn Tyr Ala Arg Leu Ala Ala Thr Lys Ala Ala Trp Asp Pro Ser His
545 550 555 560
Leu Phe Met Thr Ser Leu Gly Val Gly Ser Glu Val Trp Asp Ala Glu
565 570 575
Gly Ile Cys Arg Lys Arg Arg Gly Phe Arg Ala Lys Ala Ser Ser Leu
580 585 590
Ile Gly Met
595
<210> 74
<211> 500
<212> PRT
<213> Arthroderma benhamiae
<400> 74
Met Leu Thr Gly Val Leu Gln Asp Trp Val Trp Glu Ala Gly Glu Thr
1 5 10 15
Ala Asn Glu Ser Cys Pro Val Gly Ser Leu Arg Thr Ala Ser Ala Val
20 25 30
Asn Ser Cys His Gln Gly Arg Ile Pro Leu Phe Thr Val Gly Val Glu
35 40 45
Ser Thr Lys Gln Val Gln Glu Ala Val Arg Phe Ala Arg Lys His Asn
50 55 60
Leu Arg Leu Val Ile Arg Asn Thr Gly His Asp Leu Ala Gly Arg Ser
65 70 75 80
Ser Ala Pro Asp Ser Phe Gln Ile His Thr His His Leu Gln Glu Ile
85 90 95
Gln Phe His Ala Asp Met Arg Leu Asp Gly Ser Asn Thr Ser Leu Gly
100 105 110
Pro Ala Val Thr Val Gly Ala Gly Val Met Met Gly Asn Leu Tyr Ala
115 120 125
Gln Ala Ala Arg His Gly Tyr Met Val Leu Gly Gly Asp Cys Pro Thr
130 135 140
Val Gly Val Val Gly Gly Phe Leu Gln Gly Gly Gly Ile Ser Asp Phe
145 150 155 160
Leu Ser Leu Asn Gln Gly Phe Gly Val Asp Asn Val Leu Glu Tyr Glu
165 170 175
Val Val Thr Ala Asp Gly Glu Leu Val Val Ala Asn Ala Leu Gln Asn
180 185 190
Gln Asp Leu Phe Trp Ala Leu Arg Gly Gly Gly Gly Gly Thr Phe Gly
195 200 205
Val Val Thr Arg Ala Thr Met Arg Val Phe Pro Asp Val Pro Val Val
210 215 220
Ile Ser Glu Ile Leu Leu Glu Ala Pro Gln Ala Ile Ser Ser Ser Trp
225 230 235 240
Thr Gln Gly Leu Ser Ile Val Leu Thr Ala Leu Gln Ser Leu Asn Arg
245 250 255
Asp Asn Val Gly Gly Gln Leu Val Ile Ala Val Leu Pro Lys Leu Ala
260 265 270
Val Gln Ala Ser Ile Lys Phe Phe Phe Leu Asp Ala Thr Glu Ala Thr
275 280 285
Val Ile Asp Arg Arg Met Lys Pro Phe Leu Thr Lys Leu Ser Arg Ala
290 295 300
Asn Val Lys Tyr Thr Tyr Ser Ser Lys Asn Leu Pro His Phe Ser Ser
305 310 315 320
Asn Tyr Arg Gln Val Pro Asp Ile His Ser Asp Asn Asp Tyr Gly Val
325 330 335
Leu Gly Ser Thr Val Ala Ile Ser Gln Gln Leu Phe Asp Ser Pro Gln
340 345 350
Gly Pro Glu Lys Val Ala Thr Ala Leu Ala Asn Leu Pro Val Ser Ala
355 360 365
Gly Asp Leu Ile Phe Thr Ser Asn Leu Gly Gly Arg Val Ile Ser Asn
370 375 380
Gly Glu Leu Ala Glu Thr Ser Met His Pro Ala Trp Arg Ser Ala Ser
385 390 395 400
Gln Leu Ile Asn Tyr Val His Thr Val Glu Pro Ser Ile Glu Gly Arg
405 410 415
Ala Lys Ala Arg Glu Arg Leu Thr Asn Thr Gln Met Pro Met Leu Tyr
420 425 430
Ala Leu Asp Pro Asn Leu Lys Leu Ser Tyr Arg Asn Val Gly Asp Pro
435 440 445
Asn Glu Lys Asp Phe Gln Gln Ile Tyr Trp Gly Pro Asn Tyr Gly Arg
450 455 460
Leu Ser Asn Ile Lys Lys Lys Trp Asp Thr Asp Asp Leu Phe Phe Ser
465 470 475 480
Lys Leu Gly Val Gly Ser Glu Arg Trp Asp Ser Glu Glu Gln Leu Cys
485 490 495
Leu Leu His Ala
500
<210> 75
<211> 341
<212> PRT
<213> Paecilomyces divaricatus
<400> 75
Met Pro Thr Ser Arg Ile Ile Asp Ile Arg Arg Ser Lys Phe Glu Gly
1 5 10 15
Ser Ile Pro Asp Gln Val Val Asp Gly Leu Thr Ser Ser Pro Lys Thr
20 25 30
Leu Pro Ala Leu Leu Phe Tyr Ser Thr Glu Gly Ile Gln His Trp Asn
35 40 45
Arg His Ser His Ser Pro Asp Phe Tyr Pro Arg Leu Glu Glu Ile Arg
50 55 60
Ile Leu Lys Lys Gln Ala Ser Val Ile Ala Thr Ser Ile Ala Asp Asn
65 70 75 80
Ser Val Val Val Asp Leu Gly Ser Ala Thr Leu Asp Lys Val Thr Phe
85 90 95
Leu Leu Glu Ala Leu Glu Ala Gln Gln Lys Asn Ile Asp Tyr Tyr Ala
100 105 110
Leu Asp Leu Ser Pro Ser Glu Leu Ala Ala Thr Leu Glu Ala Val Pro
115 120 125
Ile His Gln Phe His His Val Arg Phe Ala Gly Leu His Gly Thr Phe
130 135 140
Asp Asp Gly Leu Gln Trp Leu Lys Glu Thr Pro Glu Ile Cys Asp Arg
145 150 155 160
Pro His Cys Val Leu Leu Leu Gly Leu Thr Ile Gly Asn Phe Ser Arg
165 170 175
Gln Asn Ala Ala Ser Phe Leu Arg Gly Ile Ala His His Ala Leu Ser
180 185 190
Thr Ser Pro Ala Gln Ser Ser Ile Leu Val Thr Met Asp Ser Cys Lys
195 200 205
Glu Pro Thr Lys Val Leu Arg Ala Tyr Thr Ala Asp Gly Val Val Pro
210 215 220
Phe Ala Leu Ser Ala Leu Lys Tyr Ala Asn Ser Leu Leu Arg Pro Glu
225 230 235 240
Asp Ala Glu Ile Glu Gly Gly Glu Val Phe Lys Val Asp Asp Trp Tyr
245 250 255
Phe Leu Ser Glu Trp Asn Tyr Val Leu Gly Arg His Glu Ala Ser Leu
260 265 270
Ile Pro Gln Ser Lys Asp Ile Lys Leu Gly Ala Pro Leu Glu Arg Ile
275 280 285
Val Val Arg Arg Glu Glu Lys Ile Arg Phe Gly Cys Ser Tyr Lys Tyr
290 295 300
Asn Arg Ala Glu Arg Glu Glu Leu Phe Arg Ser Ala Gly Val Ile Glu
305 310 315 320
Asp Ala Leu Trp Ser Asp Glu Gly Cys Asp Val Ala Phe Tyr Gln Leu
325 330 335
Lys Met Ile Pro Asn
340
<210> 76
<211> 339
<212> PRT
<213> Aspergillus fumigatus
<400> 76
Met Thr Ile Ser Ala Pro Pro Ile Ile Asp Ile Arg Gln Ala Gly Leu
1 5 10 15
Glu Ser Ser Ile Pro Asp Gln Val Val Glu Gly Leu Thr Lys Glu Val
20 25 30
Lys Thr Leu Pro Ala Leu Leu Phe Tyr Ser Thr Lys Gly Ile Gln His
35 40 45
Trp Asn Arg His Ser His Ala Ala Asp Phe Tyr Pro Arg His Glu Glu
50 55 60
Leu Cys Ile Leu Lys Ala Glu Ala Ser Lys Met Ala Ala Ser Ile Ala
65 70 75 80
Gln Asp Ser Leu Val Ile Asp Met Gly Ser Ala Ser Met Asp Lys Val
85 90 95
Ile Leu Leu Leu Glu Ala Leu Glu Glu Gln Lys Lys Ser Ile Thr Tyr
100 105 110
Tyr Ala Leu Asp Leu Ser Tyr Ser Glu Leu Ala Ser Asn Phe Gln Ala
115 120 125
Ile Pro Val Asp Arg Phe His Tyr Val Arg Phe Ala Ala Leu His Gly
130 135 140
Thr Phe Asp Asp Gly Leu His Trp Leu Gln Asn Ala Pro Asp Ile Arg
145 150 155 160
Asn Arg Pro Arg Cys Ile Leu Leu Phe Gly Leu Thr Ile Gly Asn Phe
165 170 175
Ser Arg Asp Asn Ala Ala Ser Phe Leu Arg Asn Ile Ala Gln Ser Ala
180 185 190
Leu Ser Thr Ser Pro Thr Gln Ser Ser Ile Ile Val Ser Leu Asp Ser
195 200 205
Cys Lys Leu Pro Thr Lys Ile Leu Arg Ala Tyr Thr Ala Asp Gly Val
210 215 220
Val Pro Phe Ala Leu Ala Ser Leu Ser Tyr Ala Asn Ser Leu Phe His
225 230 235 240
Pro Lys Gly Asp Arg Lys Ile Phe Asn Glu Glu Asp Trp Tyr Phe His
245 250 255
Ser Glu Trp Asn His Ala Leu Gly Arg His Glu Ala Ser Leu Ile Thr
260 265 270
Gln Ser Lys Asp Ile Gln Leu Gly Ala Pro Leu Glu Thr Val Ile Val
275 280 285
Arg Arg Asp Glu Lys Ile Arg Phe Gly Cys Ser Tyr Lys Tyr Asp Lys
290 295 300
Ala Glu Arg Asp Gln Leu Phe His Ser Ala Gly Leu Glu Asp Ala Ala
305 310 315 320
Val Trp Thr Ala Pro Asp Cys Asp Val Ala Phe Tyr Gln Leu Arg Leu
325 330 335
Arg Leu Asn
<210> 77
<211> 340
<212> PRT
<213> Arthroderma otae
<400> 77
Met Gly Asn Thr Asn Pro Pro His Ile Leu Asp Ile Arg Arg Ser Lys
1 5 10 15
Phe Glu Glu Ser Ile Pro Lys Gln Val Glu Ala Gly Leu Leu Ser Ser
20 25 30
Pro Lys Thr Leu Pro Ala Leu Leu Phe Tyr Ser Thr Glu Gly Ile Gln
35 40 45
His Trp Asn Arg His Ser His Ala Pro Asp Phe Tyr Pro Arg His Glu
50 55 60
Glu Ile Gln Ile Leu Lys Glu Gln Ala Thr Asp Met Ala Ala Ser Ile
65 70 75 80
Ala Asp Gly Ser Val Val Val Asp Leu Gly Ser Ala Ser Leu Asp Lys
85 90 95
Val Ile His Leu Leu Glu Ala Leu Glu Ala Ala Gln Lys Lys Val Thr
100 105 110
Tyr Tyr Ala Leu Asp Leu Ser Phe Ser Glu Leu Thr Ser Thr Leu Gln
115 120 125
Ala Ile Pro Thr Glu Gln Phe Ile His Val Gln Phe Ser Ala Leu His
130 135 140
Gly Thr Phe Glu Asp Gly Leu His Trp Leu Lys Glu Thr Pro Val Ile
145 150 155 160
Gln Asp Gln Pro His Cys Leu Leu Leu Phe Gly Leu Thr Ile Gly Asn
165 170 175
Phe Ser Arg Pro Asn Ala Ala Ala Phe Leu Arg Asn Ile Ala Ser Gln
180 185 190
Ala Leu Thr Gly Ser Pro Ser Gln Ser Ser Ile Leu Leu Thr Leu Asp
195 200 205
Ser Cys Lys Val Pro Thr Lys Val Thr Arg Ala Tyr Thr Ala Glu Gly
210 215 220
Val Val Pro Phe Ala Leu Glu Ser Leu Arg Tyr Ala Asn Thr Leu Phe
225 230 235 240
Pro Gln Asp Gly Glu Glu Arg Val Phe Asp Pro His Asp Trp His Phe
245 250 255
Leu Ser Glu Trp Asn Tyr Ile Leu Gly Arg His Glu Ala Ser Leu Ile
260 265 270
Pro Gln Ser Arg Asp Ile Lys Leu Gly Ser Pro Leu Asp Arg Ile Val
275 280 285
Val Ala Lys His Glu Lys Ile Arg Phe Gly Cys Ser Tyr Lys Tyr Asp
290 295 300
Cys Gly Glu Arg Lys Glu Leu Phe Glu Ser Ala Gly Leu His Asp Val
305 310 315 320
Lys Ile Trp Ser Lys Glu Gly Cys Asp Val Ala Phe Tyr Gln Leu Lys
325 330 335
Cys Cys Pro Asn
340
<210> 78
<211> 340
<212> PRT
<213> Trichophyton equinum
<400> 78
Met Gly Ser Ile Asn Pro Pro Gln Ile Leu Asp Ile Arg Arg Ser Lys
1 5 10 15
Phe Glu Glu Ser Ile Pro Lys Gln Val Glu Ala Gly Leu Leu Ser Ser
20 25 30
Pro Lys Thr Leu Pro Ala Leu Leu Phe Tyr Ser Thr Glu Gly Ile Gln
35 40 45
His Trp Asn Arg His Ser His Ala Ser Asp Phe Tyr Pro Arg His Glu
50 55 60
Glu Ile Gln Ile Leu Lys Asp Lys Ala Thr Asp Met Ala Ala Ser Ile
65 70 75 80
Ala Asp Gly Ser Val Val Val Asp Leu Gly Ser Ala Ser Leu Asp Lys
85 90 95
Val Ile His Leu Leu Glu Ala Leu Glu Ala Ala Gln Lys Lys Val Thr
100 105 110
Tyr Tyr Ala Leu Asp Leu Ser Phe Ser Glu Leu Asn Ser Thr Leu Lys
115 120 125
Ala Ile Pro Thr Asp Gln Phe Val His Val Gln Phe Ser Ala Leu His
130 135 140
Gly Thr Phe Asp Asp Gly Leu Gln Trp Leu Lys Glu Thr Pro Phe Ile
145 150 155 160
Arg Asp Gln Pro His Cys Leu Leu Leu Phe Gly Leu Thr Ile Gly Asn
165 170 175
Phe Ser Arg Pro Asn Ala Ala Lys Phe Leu His Asn Ile Ala Ser Tyr
180 185 190
Ala Leu Val Glu Ser Pro Ser His Ser Ser Ile Leu Leu Thr Leu Asp
195 200 205
Ser Cys Lys Val Pro Thr Lys Val Ile Arg Ala Tyr Thr Ala Glu Gly
210 215 220
Val Val Pro Phe Ala Leu Glu Ser Leu Lys Tyr Gly Asn Thr Leu Phe
225 230 235 240
His Gln Asp Val Gly Glu Asn Val Phe Asp Pro Glu Asp Trp Tyr Phe
245 250 255
Leu Ser Glu Trp Asn Tyr Val Leu Gly Arg His Glu Ala Ser Leu Val
260 265 270
Pro Arg Ser Lys Asp Ile Lys Leu Gly Arg Pro Leu Asp Lys Ile Val
275 280 285
Val Gly Lys His Glu Lys Val Arg Phe Gly Cys Ser Tyr Lys Tyr Asp
290 295 300
Ser Gly Glu Arg Lys Glu Leu Phe Glu Thr Ala Gly Leu Arg Asp Val
305 310 315 320
Lys Ser Trp Ser Lys Glu Gly Cys Asp Val Ala Phe Tyr Gln Leu Lys
325 330 335
Cys Cys Pro Asn
340
<210> 79
<211> 344
<212> PRT
<213> Epichloe brachyelytri
<400> 79
Met Ser Lys Pro Asn Val Leu Asp Ile Arg Leu Ala Thr Phe Glu Asp
1 5 10 15
Ser Ile Val Asn Leu Val Ile Asn Gly Leu Arg Lys Gln Pro Lys Thr
20 25 30
Leu Pro Ala Leu Leu Phe Tyr Ala Asn Glu Gly Leu Lys His Trp Asn
35 40 45
His His Ser His Gln Pro Glu Phe Tyr Pro Arg His Gln Glu Val Gln
50 55 60
Ile Leu Lys Lys Lys Ala Gln Glu Met Ala Ala Ser Ile Pro Met Asn
65 70 75 80
Ser Val Val Val Asp Leu Gly Ser Ala Ser Leu Asp Lys Val Ile His
85 90 95
Leu Leu Glu Ala Leu Glu Val Gln Lys Lys Asp Ile Ser Tyr Tyr Ala
100 105 110
Leu Asp Val Ser Ala Ser Gln Leu Glu Ser Thr Leu Ala Ala Ile Pro
115 120 125
Thr Gln Asn Phe Arg His Val Arg Tyr Ala Gly Leu His Gly Thr Phe
130 135 140
Asp Asp Gly Leu His Trp Leu Lys Glu Ala Pro Glu Val Arg Asp Leu
145 150 155 160
Pro His Thr Val Leu Leu Phe Gly Leu Thr Ile Gly Asn Phe Ser Arg
165 170 175
Pro Asn Ala Ala Ala Phe Leu Ser Asn Ile Gly Gln His Ala Phe Gln
180 185 190
Gly Lys Ser Gly Asp Gln Cys Ser Ile Leu Met Ser Leu Asp Ser Cys
195 200 205
Lys Val Pro Thr Gln Val Leu Arg Ala Tyr Thr Cys Glu Gly Val Val
210 215 220
Pro Phe Ala Leu Gln Ser Leu Thr Tyr Ala Asn Ser Leu Phe Gly Gly
225 230 235 240
Lys Asn Lys Arg Gln Ala Ser Gly Asp Val Gln His Lys Val Phe Asn
245 250 255
Pro Asp Glu Trp Tyr Tyr Leu Ser Glu Trp Asn Phe Val Leu Gly Arg
260 265 270
His Glu Ala Ser Leu Ile Pro Arg Ser Lys Asp Ile Glu Leu Pro Ala
275 280 285
Pro Leu Asp Gly Ile Val Val Gly Lys Asp Glu Lys Val Arg Phe Gly
290 295 300
Cys Ser Tyr Lys Tyr Asp Gln Glu Glu Arg Met Glu Leu Phe Ala Ala
305 310 315 320
Ala Gly Met Lys Asn Glu Val Thr Trp Ser Asp Glu Gly Cys Asp Ile
325 330 335
Ala Phe Tyr Glu Leu Lys Leu Ser
340
<210> 80
<211> 344
<212> PRT
<213> Neotyphodium lolii
<400> 80
Met Ser Lys Pro Asn Val Leu Asp Ile Arg Leu Ala Thr Phe Glu Asp
1 5 10 15
Ser Ile Val Asp Leu Val Ile Asn Gly Leu Arg Lys Gln Pro Lys Thr
20 25 30
Leu Pro Ala Leu Leu Phe Tyr Ala Asn Glu Gly Leu Lys His Trp Asn
35 40 45
His His Ser His Gln Pro Glu Phe Tyr Pro Arg His Gln Glu Val Gln
50 55 60
Ile Leu Lys Lys Lys Ala Gln Glu Met Ala Ala Ser Ile Pro Met Asn
65 70 75 80
Ser Val Val Val Asp Leu Gly Ser Ala Ser Leu Asp Lys Val Ile His
85 90 95
Leu Leu Glu Ala Leu Glu Val Gln Lys Lys Asn Ile Ser Tyr Tyr Ala
100 105 110
Leu Asp Val Ser Ala Ser Gln Leu Glu Ser Thr Leu Ala Ala Ile Pro
115 120 125
Thr Gln Asn Phe Arg His Val Arg Phe Ala Gly Leu His Gly Thr Phe
130 135 140
Asp Asp Gly Leu His Trp Leu Lys Glu Ala Pro Glu Ala Arg Asp Val
145 150 155 160
Pro His Thr Val Leu Leu Phe Gly Leu Thr Ile Gly Asn Phe Ser Arg
165 170 175
Pro Asn Ala Ala Ala Phe Leu Ser Asn Ile Gly Gln His Ala Phe Gln
180 185 190
Gly Lys Ser Gly Asp Gln Cys Ser Ile Leu Met Ser Leu Asp Ser Cys
195 200 205
Lys Val Pro Thr Gln Val Leu Arg Ala Tyr Thr Cys Glu Gly Val Val
210 215 220
Pro Phe Ala Leu Gln Ser Leu Thr Tyr Ala Asn Gly Leu Phe Ser Glu
225 230 235 240
Lys Asn Lys Thr Gln Ala Ser Gly Asp Val Gln His Lys Val Phe Asn
245 250 255
Leu Asp Glu Trp Tyr Tyr Leu Ser Glu Trp Asn Phe Val Leu Gly Arg
260 265 270
His Glu Ala Ser Leu Ile Pro Arg Ser Lys Asp Ile Lys Leu Leu Pro
275 280 285
Pro Leu Asp Gly Ile Leu Val Ser Lys Asp Glu Lys Val Arg Phe Gly
290 295 300
Cys Ser Tyr Lys Tyr Asp Gln Glu Glu Arg Met Glu Leu Phe Ala Ala
305 310 315 320
Ala Gly Val Lys Asn Glu Val Thr Trp Ser Asp Glu Gly Cys Asp Val
325 330 335
Ala Phe Tyr Gln Leu Lys Leu Ser
340
<210> 81
<211> 344
<212> PRT
<213> Metarhizium robertsii
<400> 81
Met Ala Ser Pro Arg Val Ile Asp Ile Arg Ser Ser His Phe Glu Asp
1 5 10 15
Leu Leu Thr Asp Gln Val Leu Ser Gly Phe Ser Asn Asp Pro Lys Thr
20 25 30
Leu Pro Ala Gln Leu Phe Tyr Thr Asn Glu Gly Leu Glu His Trp Asn
35 40 45
His His Ser Arg Gln Pro Asp Phe Tyr Pro Arg Gln Gln Glu Ile Lys
50 55 60
Ile Leu Lys Gln Arg Gly Asp Asp Met Ala Arg Ser Ile Ala Glu Asn
65 70 75 80
Ser Val Ile Leu Asp Leu Gly Ser Ala Asn Leu Glu Lys Val Thr Tyr
85 90 95
Leu Leu Glu Ala Leu Glu Ala Gln Glu Lys Asn Val Ala Tyr Phe Ala
100 105 110
Leu Asp Leu Ser Ala Ser Gln Leu Ala Ser Thr Leu Asp Ser Ile Pro
115 120 125
Thr Asp Lys Phe Arg His Val Arg Phe Thr Gly Leu His Gly Thr Phe
130 135 140
Glu Asp Gly Leu Arg Trp Ile Asn Glu Thr Pro Glu Ile Cys Asp Leu
145 150 155 160
Pro His Cys Val Leu Leu Leu Gly Leu Thr Ile Gly Asn Phe Ser Arg
165 170 175
Gln Asn Ala Ala Ala Phe Leu His Asn Ile Ala Lys Asn Ala Leu Gln
180 185 190
Gly Lys Ser Arg Ala Gln Ser Ser Ile Val Met Ser Leu Asp Ser Cys
195 200 205
Lys Val Pro Thr Gln Val Leu Arg Ala Tyr Thr Ser Asp Gly Val Val
210 215 220
Pro Phe Ala Leu Gln Ala Leu Thr Tyr Ala Asn Thr Leu Phe Gln Gly
225 230 235 240
Lys Thr Lys Asn Gly Val Asn Gly Arg Gln Ser Ser Cys Asn Leu Asp
245 250 255
Pro Glu Asp Trp Tyr Tyr Leu Ser Gln Trp Asn Phe Ala Leu Gly Arg
260 265 270
His Glu Ala Ser Leu Ile Pro Arg Ser Lys Asp Ile Asn Leu Gly Pro
275 280 285
Leu Leu Asn Asp Ile Val Val Arg Lys Asp Glu Gln Val Arg Phe Gly
290 295 300
Cys Ser Tyr Lys Tyr Asp Ala Leu Glu Arg Thr Glu Leu Phe Thr Ala
305 310 315 320
Ala Gly Leu Lys Asp Glu Thr Ala Trp Thr Gly Glu Lys Cys Asp Val
325 330 335
Ala Ile Tyr Asp Leu Lys Leu Ser
340
<210> 82
<211> 344
<212> PRT
<213> Periglandula ipomoeae
<400> 82
Met Ala Ala Pro Ser Val Ile Asp Ile Arg Ser His Leu Val Glu Asp
1 5 10 15
Ser Leu Pro Asp Gln Val Val Lys Gly Leu Gly Ser Asp Pro Lys Thr
20 25 30
Leu Pro Ala Leu Leu Phe Tyr Ser Asn Glu Gly Leu Glu Tyr Trp Asn
35 40 45
His His Ala Arg Gln Pro Asp Phe Tyr Pro Arg His Gln Glu Ile Glu
50 55 60
Ile Leu Lys Arg Lys Gly Asp Glu Ile Ala Arg Ser Val Ala Pro Asn
65 70 75 80
Ser Val Ile Leu Asp Leu Gly Ser Ala Asn Leu Glu Lys Val Thr Tyr
85 90 95
Leu Leu Glu Ala Leu Glu Ala Gln Ala Lys Asn Val Thr Tyr Phe Ala
100 105 110
Leu Asp Leu Ser Ala Pro Gln Leu Met Ser Thr Leu Lys Ala Ile Pro
115 120 125
Thr Thr Lys Phe Arg His Val Arg Phe Ala Gly Leu His Gly Thr Phe
130 135 140
Val Asp Gly Leu Arg Trp Ile Ser Glu Thr Pro Asp Ile Arg Asp Leu
145 150 155 160
Pro His Cys Val Leu Leu Phe Gly Leu Thr Ile Gly Asn Phe Ser Arg
165 170 175
Pro Asn Ala Ala Thr Phe Leu Arg Asn Ile Ala Ser Gln Ala Leu Arg
180 185 190
Gly Ala Ser Glu Asp Lys Ser Ser Ile Phe Leu Ser Leu Asp Ser Cys
195 200 205
Lys Val Pro Thr Gln Ile Leu Arg Ala Tyr Thr Ser Asp Gly Val Val
210 215 220
Pro Phe Ala Leu Gln Ser Leu Ala Tyr Ala Lys Thr Leu Phe Cys Glu
225 230 235 240
Gln Thr Gln Asn Asp Phe Asn Glu Lys Pro Ser Ser Cys His Leu Asn
245 250 255
Pro Asp Asp Trp His Tyr His Ser Glu Trp Asn Phe Val Leu Gly Arg
260 265 270
His Glu Ala Ser Leu Ile Pro Arg Leu Asn Asp Ile His Leu Gly Pro
275 280 285
Leu Leu His Asp Ile Val Val Lys Lys Asp Glu Lys Val Arg Phe Gly
290 295 300
Cys Ser Tyr Lys Tyr Asp Asp Leu Glu Arg Asp Lys Leu Phe Val Asp
305 310 315 320
Ala Gly Val Lys Asp Glu Met Ala Trp Thr Asn Glu Gly Cys Asp Ile
325 330 335
Ala Ile Tyr Glu Leu Lys Ser Met
340
<210> 83
<211> 349
<212> PRT
<213> Claviceps paspali
<400> 83
Met Lys Lys Thr Ala Ser Val Thr Asp Ile Arg Ser Ser Arg Ala Glu
1 5 10 15
Asp Ser Leu Pro Asp Gln Val Ile Lys Gly Leu Lys Ser Asp Pro Lys
20 25 30
Thr Leu Pro Ala Leu Leu Phe Tyr Ser Asp Glu Gly Leu Gly His Trp
35 40 45
Asn His His Ala Ser Gln Pro Asp Phe Tyr Pro Arg Arg Gln Glu Ile
50 55 60
Asp Ile Leu Lys Gln Arg Gly Asp Glu Met Ala Gly Ser Ile Ala Ala
65 70 75 80
Asn Ser Val Val Val Asp Leu Gly Ser Ala Asn Leu Glu Lys Val Thr
85 90 95
His Leu Leu Lys Ala Val Glu Ala Gln Glu Lys Pro Val Asp Tyr Phe
100 105 110
Ala Leu Asp Val Ala Ala Pro Gln Leu Thr Ser Thr Leu Glu Ala Met
115 120 125
Ile Ala Thr Thr Arg Phe Arg His Val Gly Leu Gly Gly Leu His Gly
130 135 140
Thr Phe Asp Asp Gly Leu Arg Trp Leu Glu Asp Thr Pro Thr Ile Glu
145 150 155 160
Asp Arg Pro Arg Cys Val Leu Leu Leu Gly Leu Thr Ile Gly Asn Phe
165 170 175
Ser Arg Arg Asn Ala Ala Ala Phe Leu Arg Ser Ile Ala Asn His Ala
180 185 190
Leu Arg Gly Pro Ala Ala Glu Ser Ser Ser Ile Leu Val Ser Ile Asp
195 200 205
Ser Cys Lys Met Pro Thr Gln Ile Leu Arg Ala Tyr Thr Ser Glu Gly
210 215 220
Val Val Pro Phe Ala Leu Gln Ser Leu Thr Cys Ala Arg Arg Leu Leu
225 230 235 240
Leu Gly Arg Gly Asp Asp Asp Asp Asp Ala Ala Leu Leu Ser Arg His
245 250 255
Leu Asn Pro Asp Asp Trp Tyr Tyr His Ser Glu Trp Asn Phe Val Leu
260 265 270
Gly Arg His Glu Ala Ser Leu Ile Pro Arg Ser Arg Asp Leu Pro Leu
275 280 285
Gly Pro Leu Leu Asp Asn Val Thr Val Gln Arg Asp Glu Lys Val Arg
290 295 300
Phe Gly Cys Ser Tyr Lys Tyr Asp Glu Ser Glu Arg Glu Thr Leu Phe
305 310 315 320
Ser Asp Ala Gly Leu Arg Cys Glu Ala Val Trp Thr Asn Gln Gly Cys
325 330 335
His Val Ala Ile Tyr Gln Leu Lys Leu Ala Ala Ser Leu
340 345
<210> 84
<211> 344
<212> PRT
<213> Claviceps purpurea
<400> 84
Met Pro Ala Leu Pro Val Ile Asp Ile Arg Ser Asn His Val Glu Asp
1 5 10 15
Ser Leu Pro Glu Gln Ile Ile Lys Gly Leu Thr Ser Gln Pro Lys Thr
20 25 30
Leu Pro Pro Leu Leu Phe Tyr Ser Asn Glu Gly Leu Glu His Trp Asn
35 40 45
His His Ser Arg Gln Pro Asp Phe Tyr Pro Arg Arg Gln Glu Ile Glu
50 55 60
Ile Leu Lys Gln Gly Gly Asn Asp Ile Ala Arg Ser Ile Ala Pro Ser
65 70 75 80
Ser Val Ile Leu Asp Leu Gly Ser Ala Asn Leu Glu Lys Val Gly Tyr
85 90 95
Leu Leu Glu Ala Leu Glu Ala Gln Glu Lys Asp Val Leu Tyr Phe Ala
100 105 110
Leu Asp Ile Ser Ala Pro Gln Leu Ala Thr Thr Leu Lys Glu Ile Pro
115 120 125
Ser Ser Asn Phe Arg His Val Arg Phe Ala Gly Leu His Gly Thr Phe
130 135 140
Glu Asp Gly Leu Arg Trp Ile Asn Glu Thr Pro Glu Ile Arg Asp Leu
145 150 155 160
Pro His Cys Val Leu Leu Leu Gly Leu Thr Ile Gly Asn Phe Ser Arg
165 170 175
Gln Asn Ala Ala Ala Phe Leu Gln Asn Ile Ala Asn His Ala Leu Thr
180 185 190
Gly Ala Ser Lys Asn Lys Ser Ser Ile Leu Leu Ser Leu Asp Ser Cys
195 200 205
Lys Val Pro Thr Lys Val Thr Arg Ala Tyr Thr Ser Asp Gly Val Val
210 215 220
Pro Phe Ala Leu Gln Ala Leu Thr Tyr Ala Lys Ala Leu Leu Cys Asp
225 230 235 240
Arg Ile Asp Asn Gly Ile Asp Glu Lys Val Leu Ser Cys Asn Leu Arg
245 250 255
Pro Glu His Trp His Tyr Leu Ser Glu Trp Asn Phe Ala Leu Gly Arg
260 265 270
His Glu Ala Ser Leu Ile Pro Arg Phe Gly Asp Val Cys Leu Gly Ser
275 280 285
Met Leu Gln Asp Ile Ile Val Lys Lys Glu Glu Lys Val Arg Phe Ala
290 295 300
Cys Ser Tyr Lys Tyr Asp Ala Lys Glu Arg Gln Lys Leu Phe Leu Asp
305 310 315 320
Ser Gly Val Asp Gln Gly Met Val Trp Thr Asn Glu Gly Cys Asp Val
325 330 335
Ala Ile Tyr Glu Leu Lys Leu Ala
340
<210> 85
<211> 355
<212> PRT
<213> Claviceps fusiformis
<400> 85
Met Pro Ala Cys Ser Val Thr Asp Ile Arg Ser His Val Val Glu Asp
1 5 10 15
Ser Leu Pro Asp Gln Val Ile Lys Gly Leu Lys Ser Ser Pro Lys Thr
20 25 30
Leu Pro Ala Leu Leu Phe Tyr Ser Asn Glu Gly Leu Asp His Trp Asn
35 40 45
His His Val Ser Gln Pro Asp Phe Tyr Pro Arg His Gln Glu Val Asp
50 55 60
Ile Leu Lys Gln Arg Gly Asp Glu Met Ala Arg Ala Ile Ala Pro Asn
65 70 75 80
Ser Val Ile Leu Asp Leu Gly Ser Ala Asn Leu Glu Lys Val Val His
85 90 95
Leu Leu Lys Ala Leu Glu Ala Gln Gly Lys Asp Val Thr Tyr Phe Ala
100 105 110
Leu Asp Ile Ser Ala Pro Gln Leu Glu Val Thr Leu Asn Glu Ile Pro
115 120 125
Thr Ser Glu Phe Arg His Val Arg Phe Ala Gly Leu His Gly Thr Phe
130 135 140
Glu Asp Gly Leu Arg Trp Ile Ser Glu Thr Pro His Ile Cys Asp Leu
145 150 155 160
Pro His Cys Val Leu Leu Leu Gly Leu Thr Ile Gly Asn Phe Ser Arg
165 170 175
Ala Ser Ala Ala Thr Phe Leu Gly Asn Ile Ala Ser Gln Ala Leu Arg
180 185 190
Gly Ala Ser Lys Asp Gln Ser Ser Ile Leu Met Ser Leu Asp Ser Cys
195 200 205
Lys Val Pro Thr Gln Ile Leu Arg Ala Tyr Thr Ser Asn Gly Val Glu
210 215 220
Pro Phe Ala Leu Gln Ser Leu Thr Phe Ala Lys Thr Leu Leu Arg Gly
225 230 235 240
Pro Met Leu His Asn Asp Ser Asp Glu Pro Leu Pro Cys Tyr Leu Gln
245 250 255
Pro Asp Asp Trp Tyr Tyr His Ser Glu Trp Asn Phe Val Leu Gly Arg
260 265 270
His Glu Ala Ser Leu Ile Pro Arg Tyr Arg Asp Val His Leu Gly Ser
275 280 285
Leu Leu Gln Asp Ile Thr Val Lys Lys Asp Glu Lys Ile Arg Phe Gly
290 295 300
Cys Ser Tyr Lys Tyr Asp Asp Met Glu Arg His Gln Leu Phe Leu Asp
305 310 315 320
Ala Gly Val Glu Gln Asp Val Ala Trp Thr Asn Glu Gly Cys Asp Val
325 330 335
Val Ile Tyr Glu Leu Lys Lys Arg Ser Asn Thr Glu Lys Leu Gly Ile
340 345 350
Asp Arg Asn
355
<210> 86
<211> 290
<212> PRT
<213> Paecilomyces divaricatus
<400> 86
Met Thr Ile Leu Val Leu Gly Gly Arg Gly Lys Thr Ala Gln Arg Leu
1 5 10 15
Ala Ser Leu Leu Asp Ala Ala Asn Ile Pro Phe Leu Leu Gly Ser Ser
20 25 30
Ser Thr Ala His Val Ser Pro Phe Arg Leu Ser Tyr Phe Asn Trp Phe
35 40 45
Glu Glu Glu Thr Tyr Gly Asn Pro Phe Val Gln Ala Ser Thr Glu Gly
50 55 60
Leu Asp Ala Val Ser Thr Val Tyr Leu Val Gly Pro Pro Val Met Asp
65 70 75 80
Met Val Pro Pro Met Leu Lys Phe Ile Asp Leu Ala Arg Ser Arg Gly
85 90 95
Val Glu Lys Phe Val Leu Leu Ser Ala Ser Thr Val Glu Lys Gly Gly
100 105 110
His Ser Met Gly Glu Val His Gly Tyr Leu Asp Ser Leu Lys Gly Val
115 120 125
Lys Tyr Val Ala Leu Arg Pro Thr Trp Phe Met Glu Asn Leu Leu Glu
130 135 140
Glu Pro His Leu Ser Trp Ile Lys Arg Gln Asn Thr Ile Tyr Ser Ala
145 150 155 160
Thr Gly Asp Gly Lys Ile Pro Phe Ile Ser Ala Asp Asp Ile Ala Arg
165 170 175
Val Ala Phe Arg Ala Leu Thr Asp Ser Asn Thr Gln Ala Thr Glu Tyr
180 185 190
Leu Ile Leu Gly Pro Glu Leu Leu Ser Tyr Gly Gln Val Ala Asp Ile
195 200 205
Leu Thr Ser Val Leu Gly Arg Lys Ile Thr His Val Ser Leu Ser Glu
210 215 220
Pro Asp Phe Thr Arg Leu Leu Val Asp Glu Val Gly Leu Pro Pro Asp
225 230 235 240
Phe Ala Ala Met Leu Ala Gly Met Glu Val Asp Val Lys Asn Gly Gly
245 250 255
Glu Asn Arg Leu Ser Asn Ser Val Lys Gln Val Thr Gly Ile Ser Pro
260 265 270
Gln Thr Phe His Asp Phe Ala Val Lys Glu Lys Gln His Trp Thr Tyr
275 280 285
Asp Gly
290
<210> 87
<211> 290
<212> PRT
<213> Aspergillus fumigatus
<400> 87
Met Thr Ile Leu Val Leu Gly Gly Arg Gly Lys Thr Ala Ser Arg Leu
1 5 10 15
Ser Leu Leu Leu Asp Asn Ala Gly Val Pro Phe Leu Val Gly Ser Ser
20 25 30
Ser Thr Ser Tyr Val Gly Pro Tyr Lys Met Thr His Phe Asp Trp Leu
35 40 45
Asn Glu Asp Thr Trp Thr Asn Val Phe Leu Arg Ala Ser Leu Asp Gly
50 55 60
Ile Asp Pro Ile Ser Ala Val Tyr Leu Val Gly Gly His Ala Pro Glu
65 70 75 80
Leu Val Asp Pro Gly Ile Arg Phe Ile Asn Val Ala Arg Ala Gln Gly
85 90 95
Val Asn Arg Phe Val Leu Leu Ser Ala Ser Asn Ile Ala Lys Gly Thr
100 105 110
His Ser Met Gly Ile Leu His Ala His Leu Asp Ser Leu Glu Asp Val
115 120 125
Gln Tyr Val Val Leu Arg Pro Thr Trp Phe Met Glu Asn Leu Leu Glu
130 135 140
Asp Pro His Val Ser Trp Ile Lys Lys Glu Asp Lys Ile Tyr Ser Ala
145 150 155 160
Thr Gly Asp Gly Lys Ile Pro Phe Ile Ser Ala Asp Asp Ile Ala Arg
165 170 175
Val Ala Phe Ser Val Leu Thr Glu Trp Lys Ser Gln Arg Ala Gln Glu
180 185 190
Tyr Phe Val Leu Gly Pro Glu Leu Leu Ser Tyr Asp Gln Val Ala Asp
195 200 205
Ile Leu Thr Thr Val Leu Gly Arg Lys Ile Thr His Val Ser Leu Ala
210 215 220
Glu Ala Asp Leu Ala Arg Leu Leu Arg Asp Asp Val Gly Leu Pro Pro
225 230 235 240
Asp Phe Ala Ala Met Leu Ala Ser Met Glu Thr Asp Val Lys His Gly
245 250 255
Thr Glu Val Arg Asn Ser His Asp Val Lys Lys Val Thr Gly Ser Leu
260 265 270
Pro Cys Ser Phe Leu Asp Phe Ala Glu Gln Glu Lys Ala Arg Trp Met
275 280 285
Arg His
290
<210> 88
<211> 293
<212> PRT
<213> Paracoccidioides brasiliensis
<400> 88
Met Thr Ile Leu Ile Leu Gly Gly Arg Gly Lys Thr Ser Ser Arg Leu
1 5 10 15
Ala Ser Ile Leu Ser Thr Arg Ala Pro Glu Ile Pro Phe Ile Val Ala
20 25 30
Ser Arg Thr Thr Ser Pro Pro Ser Pro His Lys Gln Ala Ile Phe Asp
35 40 45
Trp Leu Asp Glu Ser Thr Trp Gly Gln Pro Phe Glu Met Ala Ser Thr
50 55 60
Ser Ser Ala Phe Thr Leu Gly Pro Ile Ser Ala Val Tyr Ile Val Ser
65 70 75 80
Pro Pro Val Val Asp Leu Phe Pro Leu Ala Lys Arg Phe Ile Asp Phe
85 90 95
Ala Arg Glu Lys Gly Val Lys Arg Phe Val Leu Leu Ser Ala Ser Thr
100 105 110
Phe Glu Lys Gly Gly Pro Leu Met Gly Lys Val His Glu Tyr Leu Ala
115 120 125
Gly Leu Gly Glu Glu Ile Ala Trp Thr Val Leu Arg Pro Thr Trp Phe
130 135 140
Met Gln Asn Val Ser Glu Ala Gln Phe Leu Pro Phe Ile Lys Gly Glu
145 150 155 160
Asp Lys Ile Tyr Ser Ser Thr Gly Glu Gly Lys Ile Pro Phe Val Ser
165 170 175
Thr Asp Asp Ile Ala Arg Val Ala Phe His Ala Leu Thr Asp Glu Arg
180 185 190
Pro His Asn Thr Asp His Leu Ile Leu Gly Pro Glu Leu Leu Ser Tyr
195 200 205
Gly Asp Ile Ala Asp Ile Leu Ser Ser Val Leu Gly Arg Lys Ile Thr
210 215 220
His Val Ser Leu Ser Glu Ala Asp Tyr Val Ala Trp Leu Met Ser Ala
225 230 235 240
Thr Gly Val Ser Glu Asn Tyr Ala Lys Met Leu Ala Gly Leu Glu Ile
245 250 255
Pro Ile Arg Asn Arg Glu Leu Glu Glu Val Asn Asp Val Val Glu Arg
260 265 270
Val Thr Gly Lys Ala Pro Met Thr Phe Ser Glu Phe Ala Glu Val Ala
275 280 285
Lys Glu Cys Trp Leu
290
<210> 89
<211> 289
<212> PRT
<213> Claviceps fusiformis
<400> 89
Met Thr Ile Leu Leu Thr Gly Gly Ser Gly Lys Thr Ala Gly His Ile
1 5 10 15
Ala Asn Leu Leu Lys Glu Ala Lys Leu Pro Phe Ile Val Gly Ser Arg
20 25 30
Ser Ser Asn Pro His Thr Val Glu Arg His Arg Thr Phe Asp Trp Leu
35 40 45
Asp Glu Ala Thr Phe Asn Asn Val Leu Ser Val Asp Glu Gly Met Glu
50 55 60
Pro Val Ser Val Val Trp Leu Val Ser Pro Pro Ile Leu Asp Leu Ala
65 70 75 80
Pro Pro Val Ile Arg Phe Ile Asp Phe Ala Ser Ser Arg Gly Val Lys
85 90 95
Arg Phe Val Leu Leu Ser Ala Ser Thr Val Glu Lys Gly Gly Pro Ala
100 105 110
Met Gly Leu Ile His Ala His Leu Asp Thr Ile Glu Gly Val Ser Tyr
115 120 125
Thr Val Leu Arg Pro Ser Trp Phe Met Glu Asn Phe Ser Thr Arg Gly
130 135 140
Glu Phe Pro Cys Asp Thr Ile Arg Glu Glu Asp Thr Ile Tyr Ser Ala
145 150 155 160
Ala Lys Asp Gly Lys Ile Pro Phe Ile Ser Val Ala Asp Ile Ala Arg
165 170 175
Val Ala Leu Arg Ala Leu Thr Ala Pro Ala Leu His Asn Lys Asp His
180 185 190
Val Leu Leu Gly Pro Glu Leu Leu Thr Tyr Asp Asp Val Ala Glu Ile
195 200 205
Leu Thr Arg Val Val Gly Arg Asn Ile His His Val Arg Leu Thr Glu
210 215 220
Ser Glu Leu Ala Ala Lys Leu Gln Glu Arg Gly Met Pro Ala Asp Glu
225 230 235 240
Ala Ala Met His Ala Ser Leu Asp Ser Ile Val Glu Ala Gly Ala Glu
245 250 255
Glu Lys Leu Asn Thr Glu Val Lys Asp Leu Thr Gly Glu Glu Pro Arg
260 265 270
His Phe Ala Asp Phe Val Ser Asp Asn Lys Asn Val Trp Leu Met Arg
275 280 285
Asp
<210> 90
<211> 286
<212> PRT
<213> Epichloe glyceriae
<400> 90
Met Thr Ile Leu Leu Thr Gly Gly Arg Gly Lys Thr Ala Ser His Ile
1 5 10 15
Ala Ser Leu Leu Gln Ala Ala Lys Val Pro Phe Ile Val Ala Ser Arg
20 25 30
Ser Ser Asn Ser Ser Ser Pro Tyr Tyr Gln Asn Cys Phe Asp Trp Leu
35 40 45
Asp Glu Lys Thr Tyr Gly Asp Ala Leu Thr Pro Lys Glu Gly Met Gln
50 55 60
Pro Ile Ser Thr Val Trp Leu Val Pro Pro Pro Ile Phe Asp Leu Ala
65 70 75 80
Pro Pro Met Ile Lys Phe Val Asp Phe Ala Ser Arg Arg Gly Val Glu
85 90 95
Arg Phe Val Leu Leu Ser Ala Ser Thr Ile Glu Lys Gly Gly Pro Ala
100 105 110
Met Gly Gln Val His Glu Tyr Leu Ala Ser Leu Gly Gly Ile Glu Tyr
115 120 125
Ala Val Leu Arg Pro Thr Trp Phe Met Glu Asn Phe Ser Tyr Pro Gln
130 135 140
Glu Leu Gln Arg Leu Ala Ile Lys Asn Glu Asn Lys Ile Tyr Ser Ala
145 150 155 160
Ala Gly Asp Gly Glu Leu Pro Phe Val Ser Val Ala Asp Ile Ala Arg
165 170 175
Val Ala Phe Arg Thr Leu Thr Asp Glu Lys Ser His Asn Thr Asp Tyr
180 185 190
Val Leu Leu Gly Pro Glu Leu Met Thr Tyr Asp Gln Val Ala Glu Thr
195 200 205
Leu Ser Thr Val Leu Gly Arg Thr Ile Thr His Val Lys Leu Thr Lys
210 215 220
Asp Glu Leu Ala Lys Arg Leu Glu Asn Gly Gly Met Thr Ala Glu Asp
225 230 235 240
Ala Glu Met Leu Ala Gly Met Asp Thr Ser Ile Ser Asp Gly Ala Glu
245 250 255
Asp Arg Leu Asn Asn Val Val Lys His Val Thr Gly Ala Asp Pro Arg
260 265 270
Thr Phe Leu Asp Phe Ala Thr Glu Gln Lys Ala Thr Trp Gly
275 280 285
<210> 91
<211> 309
<212> PRT
<213> Neotyphodium lolii
<400> 91
Met Val Gln Ile Tyr Leu Pro Arg Leu Ser Met Arg Leu Gly Tyr His
1 5 10 15
Asp Lys Asn Asn Lys Met Thr Ile Leu Leu Thr Gly Gly Arg Gly Lys
20 25 30
Thr Ala Ser His Ile Ala Ser Leu Leu Gln Ala Ala Lys Val Pro Phe
35 40 45
Ile Val Ala Ser Arg Ser Ser Asp Pro Ser Ser Ser Ser Pro Tyr Tyr
50 55 60
Gln Asn Cys Phe Asp Trp Leu Asp Glu Lys Thr Tyr Gly Asp Val Leu
65 70 75 80
Thr Ser Lys Asp Ser Met Gln Pro Ile Ser Thr Ile Trp Leu Val Pro
85 90 95
Pro Pro Ile Phe Asp Leu Ala Pro Leu Met Ile Lys Phe Val Asp Phe
100 105 110
Ala Ser Arg Lys Gly Val Lys Arg Phe Val Leu Leu Ser Ala Ser Thr
115 120 125
Ile Lys Lys Gly Gly Pro Ala Met Gly Gln Val His Glu Tyr Leu Ala
130 135 140
Ser Leu Gly Gly Ile Glu Tyr Ala Val Leu Arg Pro Thr Trp Phe Met
145 150 155 160
Glu Asn Phe Ser Tyr Pro Gln Glu Leu Gln Arg Leu Ala Ile Lys Asn
165 170 175
Glu Asn Lys Ile Tyr Ser Ala Ala Gly Asp Gly Lys Leu Pro Phe Val
180 185 190
Ser Val Ala Asp Ile Ala Arg Val Ala Phe Arg Thr Leu Thr Asp Glu
195 200 205
Lys Ser His Asn Thr Asp Tyr Val Leu Leu Gly Pro Glu Leu Ile Thr
210 215 220
Tyr Asp Gln Val Ala Glu Thr Leu Ser Thr Val Leu Gly Arg Thr Ile
225 230 235 240
Thr His Ile Lys Leu Thr Glu Glu Glu Leu Val Lys Arg Leu Glu Asn
245 250 255
Ser Gly Met Pro Ala Glu Asp Ala Lys Met Leu Ala Gly Met Asp Thr
260 265 270
Ser Ile Ser Asp Gly Ala Glu Asp Arg Leu Asn Asn Val Val Lys His
275 280 285
Val Thr Gly Ala Asp Pro Arg Thr Phe Leu Asp Phe Ala Thr His Gln
290 295 300
Lys Ala Thr Trp Gly
305
<210> 92
<211> 288
<212> PRT
<213> Claviceps paspali
<400> 92
Met Thr Ile Leu Leu Thr Gly Gly Asn Gly Lys Thr Thr Arg Gln Ile
1 5 10 15
Ala Arg Leu Phe Glu Glu Ala Lys Leu Pro Phe Val Thr Ala Ser Arg
20 25 30
Ser Pro Ser His Pro Ala Ala Lys Val His Arg Lys Phe Asp Trp Leu
35 40 45
Asp Glu Ala Thr Phe Ala Asn Ala Leu Ser Ser Asp Asp Gly Met Lys
50 55 60
Pro Val Ser Ala Ile Trp Leu Val Ala Pro Pro Val Ala Asp Pro Ala
65 70 75 80
Pro Pro Val Ile Lys Phe Ile Asp Leu Ala Arg Glu Lys Gly Val Lys
85 90 95
Arg Phe Val Leu Leu Ser Ala Ser Ile Ile Glu Lys Gly Gly Pro Ala
100 105 110
Met Gly Ser Ile His Ala His Leu Asp Ser Leu Ala Gly Ile Ser Tyr
115 120 125
Ala Val Leu Arg Pro Thr Trp Phe Met Glu Asn Phe Ser Thr Ala Gly
130 135 140
Glu Phe Gln Phe Glu Thr Met Ala Lys Glu Asn Asn Ile Tyr Ser Ala
145 150 155 160
Ala Glu Asp Gly Lys Ile Pro Phe Ile Ser Thr Val Asp Ile Ala Arg
165 170 175
Val Ala Phe Arg Ala Leu Thr Ala Asp Ser Ile Asp Ala Lys Asp Leu
180 185 190
Val Leu Val Gly Pro Glu Leu Leu Thr Tyr Asp Asp Val Ala Lys Thr
195 200 205
Leu Ser Lys Val Leu Gly Arg Asp Ile Thr His Val Arg Val Thr Glu
210 215 220
Ala Ala Leu Ala Glu Lys Arg Arg Glu Ser Gly Val Pro Ala Asp Ile
225 230 235 240
Ala Asp Met Ile Ala Ser Met Asp Leu Ile Val Arg Ser Gly Ala Glu
245 250 255
Asp Arg Met Asn Thr Ala Val Lys Asp Phe Thr Gly Val Glu Pro Arg
260 265 270
Ser Phe Ala Glu Phe Ala Ser Lys Glu Arg Glu Ala Trp Leu Ser Thr
275 280 285
<210> 93
<211> 290
<212> PRT
<213> Claviceps purpurea
<400> 93
Met Thr Val Leu Leu Thr Gly Gly Thr Gly Arg Thr Ala Lys His Ile
1 5 10 15
Ala Gly Ile Phe Arg Gln Thr Asn Val Pro Phe Leu Val Ala Ser Arg
20 25 30
Ser Ser Ser Ala Gly Thr Ala Glu Asn His Arg Lys Phe Asp Trp Leu
35 40 45
Asp Glu Glu Thr Phe Pro Asn Ala Leu Ser Val Asp Gln Gly Met Lys
50 55 60
Pro Ile Ser Val Val Trp Leu Cys Pro Pro Pro Leu Tyr Asp Leu Ala
65 70 75 80
Thr Pro Val Ile Lys Phe Ile Asp Phe Ala Val Ser Gln Asn Val Lys
85 90 95
Lys Phe Val Leu Leu Ser Ala Ser Val Ile Gln Lys Gly Gly Pro Ala
100 105 110
Met Gly Lys Ile His Gly His Leu Asp Ser Ile Lys Asp Val Thr Tyr
115 120 125
Thr Val Leu Arg Pro Thr Trp Phe Met Glu Asn Phe Ser Thr Lys Gly
130 135 140
Glu Ile Gln Cys Glu Ala Ile Arg Arg Asp Ser Thr Val Tyr Ser Ala
145 150 155 160
Thr Glu Asn Gly Lys Ile Pro Phe Ile Ser Val Val Asp Ile Ala Arg
165 170 175
Val Ala Ala Cys Ala Leu Thr Ala Glu Thr Leu Lys Asn Ser Asp His
180 185 190
Ile Leu Gln Gly Pro Asp Leu Leu Thr Tyr Asp Glu Val Ala Gln Ala
195 200 205
Leu Thr Gly Val Leu Gly Arg Lys Ile Thr His Thr Lys Met Thr Glu
210 215 220
Gly Glu Leu Ala Glu Lys Leu Met Glu Glu Gly Val Thr Pro Glu Glu
225 230 235 240
Ala Tyr Met His Ala Ala Met Asp Ser Met Ile Lys Ser Gly Ser Glu
245 250 255
Glu Arg Val Val Ser Asp Glu Val Lys Glu Trp Thr Gly Val Lys Pro
260 265 270
Arg Gly Phe Ile Asn Phe Ala Leu Ser Glu Lys Ala Ala Trp Arg Ala
275 280 285
Arg Lys
290
<210> 94
<211> 308
<212> PRT
<213> Metarhizium robertsii
<400> 94
Met Thr Ile Leu Leu Thr Gly Gly Arg Gly Lys Thr Ala Ser His Ile
1 5 10 15
Ala Ala Leu Leu Asp Ala Ala Asn Leu Pro Phe Ala Ile Ala Ser Arg
20 25 30
Ser Cys Ser His Asp Ser Lys Tyr Arg Gln Leu Arg Phe Asp Trp Leu
35 40 45
Asp Asp Thr Thr Tyr Asp Asn Val Leu Ser Pro Lys Asp Gly Met Gln
50 55 60
Pro Ile Ser Ala Val Trp Leu Val Pro Pro Pro Ile Ala Asp Leu Ala
65 70 75 80
Pro Pro Val Asn Ala Phe Ile Asn His Ala Arg Leu Lys Asp Val Lys
85 90 95
Arg Phe Val Leu Leu Ser Ala Ser Met Leu Glu Arg Gly Ala Pro Ala
100 105 110
Met Gly Gln Ile His Glu His Leu Ala Ser Met Glu Gly Ile His Tyr
115 120 125
Thr Val Leu Arg Pro Thr Trp Phe Met Glu Asn Phe Ser Ser Pro His
130 135 140
Glu Phe Gln Cys Lys Ala Ile Arg Lys Glu Asp Lys Ile Tyr Ser Ala
145 150 155 160
Ala Glu Asn Gly Lys Val Pro Phe Ile Ser Val Val Asp Ile Ala Arg
165 170 175
Val Ala Phe Arg Ala Leu Lys Gly Asp Val Glu Glu Asn Ile Asp Pro
180 185 190
Ile Leu Leu Gly Pro Glu Leu Leu Thr Tyr Asp Asp Val Ser Thr Cys
195 200 205
Lys Gly Leu Arg Asp Ala Ser Pro Asp Thr Asp Lys Arg Val Lys Val
210 215 220
Ala Glu Thr Leu Ser Lys Gln Leu Asn Arg Lys Ile Asn His Val Arg
225 230 235 240
Leu Glu Glu Ser Glu Leu Ala Glu Lys Met His Glu Tyr Gly Met Pro
245 250 255
Leu Glu Asp Ala Gln Met His Ala Ser Met Asp Leu Ile Val Arg Ala
260 265 270
Gly Gly Glu Glu Arg Leu Asn Lys Thr Val Leu Glu Leu Thr Gly Thr
275 280 285
Thr Pro Arg Thr Phe Gln Glu Phe Ala Ser Ser Glu Lys Asp Ile Trp
290 295 300
Leu Leu Lys Asn
305
<210> 95
<211> 289
<212> PRT
<213> Paecilomyces divaricatus
<400> 95
Met Thr Val Thr Lys Thr Ser Lys Pro Val Leu Arg Arg Leu Pro Ile
1 5 10 15
Ser Ala Gly Ala Asp Ala Ile Phe Gln Val Leu Gln Glu Asp Gly Val
20 25 30
Leu Ile Ile Glu Gly Phe Leu Thr Pro Glu Gln Val Lys Lys Phe Asn
35 40 45
Ala Glu Val Asp Pro His Leu Lys Lys Trp Glu Leu Gly Gln Gly Ser
50 55 60
Met Gln Glu Gly Tyr Leu Ala Asn Met Lys Gln Leu Ser Ser Leu Pro
65 70 75 80
Leu Phe Ser Lys Thr Phe Arg Asp Asp Leu Met Asn His Asp Leu Leu
85 90 95
His Gly Ile Cys Lys Lys Ala Phe Gly Pro Asp Ser Gly Asp Tyr Trp
100 105 110
Leu Thr Thr Ser Ser Val Leu Glu Thr Asp Pro Gly Tyr Pro Gly Gln
115 120 125
Asp Leu His Arg Glu His Asp Gly Ile Pro Ile Cys Thr Thr Leu Gly
130 135 140
Arg Asp Ser Pro Glu Ala Met Leu Asn Phe Leu Thr Ala Leu Thr Asp
145 150 155 160
Phe Thr Glu Glu Asn Gly Ala Thr Arg Val Leu Pro Gly Ser His Leu
165 170 175
Trp Glu Asp Tyr Ser Lys Ser Val Ser Pro Asp Gln Ala Ile Pro Ala
180 185 190
Glu Met Asn Pro Gly Asp Ala Val Leu Phe Ser Gly Lys Thr Leu His
195 200 205
Gly Ala Gly Lys Asn Gln Ser Lys Asp Phe Leu Arg Arg Gly Phe Pro
210 215 220
Leu Ile Met Gln Ser Cys Gln Phe Thr Pro Val Glu Ala Ser Val Ala
225 230 235 240
Ile Pro Arg Ser Leu Val Glu Thr Met Thr Pro Leu Ala Gln Lys Met
245 250 255
Val Gly Trp Arg Ser Val Ser Ala Lys Gly Val Glu Ile Trp Thr Tyr
260 265 270
Asp Leu Lys Asp Leu Ala Lys Gly Leu Lys Leu Lys Ala Ile Ala Ala
275 280 285
Glu
<210> 96
<211> 298
<212> PRT
<213> Botryotinia fuckeliana
<400> 96
Met Thr Val Ser Val Pro Thr Pro Phe Arg Arg Ile Ser Ala Lys Ala
1 5 10 15
Pro Val Glu Glu Ile Leu Gln Met Phe Arg Glu Asp Gly Gly Ile Val
20 25 30
Val Glu Asp Phe Phe Thr Gln Glu Gln Met Ala Asn Ile Asn Lys Glu
35 40 45
Val Asp Pro Leu Leu Ala Lys Ile Lys Cys Gly Thr His Arg Asp Val
50 55 60
Gly Glu Glu Glu Trp Ile Thr Gln Phe His Gly Ser Asn Thr Arg Arg
65 70 75 80
Leu Ser Asn Leu Pro Thr Ile Ser Lys Thr Phe Arg Glu Glu Val Leu
85 90 95
Asn Tyr Asp Leu Leu His Gln Ile Cys Glu Ala Ile Phe Arg Lys Glu
100 105 110
Ser Gly Asp Tyr Trp Met Cys Thr Ala Gln Val Ile Glu Ile Gly Pro
115 120 125
Gln Ser Lys Ala Gln Glu Leu His Arg Asp Thr Ala Glu Phe Pro Gly
130 135 140
Leu Thr Lys Leu Gly Pro Arg Met Asp Glu Ile Gln Ile Ser Phe Phe
145 150 155 160
Thr Ala Leu Thr Asp Phe Thr Ala Glu Asn Gly Ala Thr Arg Gly Ile
165 170 175
Pro Lys Ser His Leu Met Glu Asp Tyr Glu Asp Trp Gly Arg Glu Glu
180 185 190
Asp Ala Val Pro Ala Val Met Lys Thr Gly Asp Val Phe Phe Phe Thr
195 200 205
Gly Arg Leu Cys His Gly Gly Gly Glu Asn Arg Thr Glu Asp Phe Trp
210 215 220
Arg Arg Gly Ile Ser Leu Cys Phe Gln Ala Ser Tyr Leu Thr Pro Glu
225 230 235 240
Glu Cys Tyr Pro Leu Met Ile Pro Arg Asp Val Ile Glu Ser Met Thr
245 250 255
Pro Leu Ala Gln Lys Met Cys Ala Trp Arg Ser Gln Tyr Pro Thr Leu
260 265 270
Gly Gly Gly Leu Trp Gln His Asn Tyr Thr Glu Val Ala Asp Gln Ile
275 280 285
Gly Leu Lys Ser Asn Gln Pro Leu Lys Leu
290 295
<210> 97
<211> 613
<212> PRT
<213> Chaetomium globosum
<400> 97
Met Pro Ser Phe Val Thr Pro Ala Ile Leu Ile Asn Asn Ser Thr Ser
1 5 10 15
Tyr Pro Leu Leu Leu Ile Thr Ala Leu Glu Asp Thr Gly Ile Leu Asp
20 25 30
Ser Leu Ile Val Thr Asp Glu Thr Ala Lys Gln Ala Ile Glu Arg Ala
35 40 45
Lys Ala Tyr Leu Leu Val Tyr Ser Thr Ile Ser Asn Cys Val Thr Phe
50 55 60
Ala Val Gly Pro Arg Leu Ile Asp Ser Glu His Ala Ala Asp Asp Glu
65 70 75 80
Asp Asp Glu Asp Asp Lys Thr Gly Asn Gly Asn Arg Gly Arg Asp Gly
85 90 95
His Thr Ala Ala Ser Thr Pro Asp Val Glu Ala Asn Glu Glu Thr Arg
100 105 110
Leu Leu Asp Ser Gln Leu Pro Asn Ser Pro His Pro Leu Arg Arg Ser
115 120 125
Ser Phe Ser Phe Ser Leu Pro Ser Lys Ser Gly Lys Pro Thr Ser Lys
130 135 140
Pro Asp His Arg Arg Pro Trp Phe Met Ser Pro Gln Arg Trp Asn Arg
145 150 155 160
Leu Ser Ser Gln Thr Lys Trp Trp Phe Leu Phe Ile Leu Asp Phe Phe
165 170 175
Asn Ala Pro Leu Leu Gly Ala Ile Val Gly Ala Val Val Gly Leu Val
180 185 190
Pro Pro Phe His Arg Ala Phe Phe Asn Ser Ser Asp Asp Gly Gly Ile
195 200 205
Phe Thr Ala Trp Leu Thr Ser Ala Leu Lys Gln Ile Gly Gly Leu Phe
210 215 220
Val Ser Leu Pro Val Val Val Ala Gly Ile Thr Leu Phe Cys Ser Thr
225 230 235 240
Lys Glu Ala Arg Ala Asn Asn Glu Ser Gly Met Asn Leu Pro Leu Gly
245 250 255
Thr Thr Leu Tyr Ile Leu Phe Val Arg Phe Ile Ala Trp Pro Ala Val
260 265 270
Ser Ile Ser Val Ile Tyr Val Leu Ala Ala Lys Thr Ser Leu Leu Gly
275 280 285
Ser Asp Pro Val Leu Trp Phe Cys Leu Met Met Met Pro Glu Trp Thr
290 295 300
Lys Arg His Glu Ala His His Pro Arg Phe Arg Trp Leu Arg Glu Pro
305 310 315 320
Arg Arg Leu Leu Ser Leu His Gly Lys Met Pro Ala Thr Pro Lys Leu
325 330 335
Gln Gln Val Lys Pro Ser Glu Gly Arg Asp Ala Val Phe Arg Ala Leu
340 345 350
Arg Glu Asp Gly Ala Val Ile Ile Lys Gly Leu Phe Thr Lys Asp Gln
355 360 365
Val Arg Arg Leu Asn His Glu Val Gln Pro Ala Met Asp Lys Ile Gly
370 375 380
Val Gly Ser Lys Gln Ser Glu Glu Trp Leu Lys Asp Phe His Gly Asp
385 390 395 400
Lys Thr Lys Arg Leu Asn Gly Val Val Ala Leu Ser Glu Thr Phe Arg
405 410 415
His Glu Ile Leu Glu Asn Asp Leu Ile His Glu Leu Cys Glu Glu Ile
420 425 430
Tyr Leu Gln Asp Ala Gly Gly Tyr Trp Met Asn Ser Ala Gln Val Ile
435 440 445
Asp Ile Gly Pro Gly Ser Lys Ala Gln Pro Leu His Arg Asp Gln Trp
450 455 460
Gln Phe Pro Ile Phe Thr Tyr Ser Gly Pro Asp Ala Pro Glu Ala Ser
465 470 475 480
Val Asn Phe Val Val Ala Leu Thr Glu Phe Thr Asp Glu Asn Gly Ala
485 490 495
Thr Arg Val Ile Pro Gly Ser His Arg Trp Gln Asp Leu Gln Gln Asn
500 505 510
Gly Thr Pro Glu Asp Thr Ile Pro Ala Glu Met Glu Val Gly Asp Ala
515 520 525
Cys Phe Ile Thr Gly Lys Val Val His Gly Gly Gly Ala Asn Arg Thr
530 535 540
Lys Asp Phe Thr Arg Arg Ala Ile Thr Leu Val Phe Gln Cys Ser Tyr
545 550 555 560
Leu Thr Pro Glu Glu Ala Tyr Pro Phe Ile Val Ser Lys Glu Ile Ala
565 570 575
Lys Thr Leu Ser Pro Arg Gly Gln Arg Met Ile Gly Phe Arg Ser Gln
580 585 590
Phe Leu Lys Asp Ser Pro Gly Val Trp Lys Arg Asp Tyr Gly Glu Val
595 600 605
Asp Glu Ala Tyr Cys
610
<210> 98
<211> 286
<212> PRT
<213> Aspergillus terreus
<400> 98
Met Thr Gly Gln Ala Glu Ala Ile Arg Arg Val His Pro Thr Val Ser
1 5 10 15
Pro Lys Gln Ala Ala Gln Met Leu Gln Glu Asp Gly Val Ile Ile Leu
20 25 30
Lys Ser Phe Leu Ala Pro Asp Val Met Gln Arg Phe Gln Ala Glu Val
35 40 45
Asp Glu Asp Val Glu Lys Thr Ser Thr Gly Ala Arg Met Lys Ala Tyr
50 55 60
Lys Leu Val Asn Asp Lys Thr Lys His Met Ala Asp Leu Ile Val Arg
65 70 75 80
Ser Glu Val Phe Arg Ser Asp Ile Leu Thr His Pro Leu Tyr His Ala
85 90 95
Ile Ala Asp Glu Leu Phe Arg Ala Asp Tyr Gly Asp His Trp Leu Asn
100 105 110
Ala Ser Ala Val Leu Gln Leu Met Pro Gly Ala Pro Ala Gln Gln Leu
115 120 125
His Arg Asp Glu Glu Ile Phe Ala Ala Ser Lys Phe Arg Ser Pro Thr
130 135 140
Asp Pro Gln Leu Ser Leu Ser Cys Leu Val Ala Leu Thr Glu Phe Thr
145 150 155 160
Glu Glu Asn Gly Ala Thr Arg Leu Ile Pro Gly Ser His Leu Trp Asp
165 170 175
Ser Ala His Pro Ala Pro Ser Pro Asp Gln Thr Val Pro Ala Ile Met
180 185 190
Gln Pro Gly Glu Ala Ile Leu Phe Leu Gly Ser Leu Phe His Gly Gly
195 200 205
Gly Glu Asn Arg Thr Glu Asn Val Arg Arg Gly Leu Gly Met Ser Leu
210 215 220
Ile Pro Cys Gln Phe Thr Pro Tyr Ser Ser His Met His Val Pro Arg
225 230 235 240
Thr Ile Ile Glu Thr Met Thr Pro Leu Ala Gln Lys Leu Val Gly Trp
245 250 255
Arg Thr Val Glu Ser His Arg Gln Tyr Pro Phe Trp Gln Gly Gly Asp
260 265 270
Arg Arg Leu Glu Asp Val Leu Gly Leu Ala Ser Arg Glu Ala
275 280 285
<210> 99
<211> 297
<212> PRT
<213> Neosartorya fischeri
<400> 99
Met Thr Val Pro Asp Asn Ser Ser Lys Pro Gln Ile Gln Arg Val Pro
1 5 10 15
Ala Asp Ala Asp Pro Asn Ala Ile Arg Gln Leu Leu Glu Glu Asp Gly
20 25 30
Ala Ala Ile Ile Glu Gly Val Phe Ser Glu Asp Ile Ile Gln Ser Phe
35 40 45
Asn Gln Asp Leu Asn Pro Ala Ala Thr Ala Ala Glu Met Ala Ala Lys
50 55 60
Thr Asn Tyr Glu Phe Gln Phe Pro Gln Gln Ser Lys Tyr Val Phe Asn
65 70 75 80
Leu Pro Gly Lys Ser Ala Thr Phe Arg Asn Asp Ile Leu Asn Ser Pro
85 90 95
Val Leu His Gly Leu Ala Lys Ser Val Phe Ala Glu Thr Gly Asp Tyr
100 105 110
Trp Leu Thr Ala Ser Phe Leu Arg Glu Leu His Ala Gly His Asn Pro
115 120 125
Gln Glu Leu His Arg Asp Glu Ser Thr His Pro Leu Leu Arg Ser Leu
130 135 140
Lys Leu Gly Ala Pro Pro Leu Ala Ile Ser Phe Ile Leu Ala Leu Thr
145 150 155 160
Asp Phe Thr Lys Asp Asn Gly Gly Thr Arg Val Ile Leu Gly Ser His
165 170 175
Lys Trp Pro Glu Ile Gly Asn Pro Arg Pro Asp Gln Thr Val Ser Thr
180 185 190
Glu Met Lys Ala Gly Asp Val Leu Val Leu Ala Gln Gly Met Val His
195 200 205
Gly Gly Gly Ala His Pro Glu Asp Leu Pro Glu Ala Arg Arg Ala Val
210 215 220
Leu Val Phe Phe Ser Ser Cys Gln Leu Thr Pro Phe Glu Thr Phe Met
225 230 235 240
Thr Met Pro Arg Glu Ile Val Glu Thr Met Thr Pro Leu Ala Gln Gln
245 250 255
Met Ile Gly Trp Arg Ser Ala Lys Pro Gly Pro Pro Asn Val Ser Gly
260 265 270
Leu His Thr Ser Asp Ser Lys Leu Leu Glu Asp Ala Leu Gln Leu Arg
275 280 285
Ala Asn Gln Ser Gln Gly Lys Gly Leu
290 295
<210> 100
<211> 298
<212> PRT
<213> Uncinocarpus reesii
<400> 100
Met Val Ala Ile Gly Asp Ser Lys Pro Gln Ile Lys Arg Phe Pro Val
1 5 10 15
Ser Glu Asn Pro Glu Thr Ile Trp Lys Val Val Glu Glu Asp Gly Ala
20 25 30
Ala Ile Val Glu Gly Trp Leu Ser Pro Asp Val Val Gln Arg Phe Asn
35 40 45
Gln Asp Leu Asp Val Arg Ser Ala Lys Thr Pro Gly Gly Thr Met Asn
50 55 60
Glu Glu Phe Tyr Thr Met Pro Leu Pro Lys Thr Thr Lys Trp Met Asn
65 70 75 80
Asp Leu Pro Ala Thr Cys Lys Thr Phe Arg His Asp Ile Leu Asn Asn
85 90 95
Pro Ile Leu His Asp Ile Cys Lys Ile Ala Phe Lys Glu His Gly Asp
100 105 110
Tyr Trp Leu Leu Asn Gly Met Ala Met Glu Met Ala Pro Gly Asn Pro
115 120 125
Ile Gln Gln Ile His Arg Asp Gln Gly Thr His Pro Ile Leu Lys Tyr
130 135 140
Ile Lys Pro Gly Ser Pro Val Pro Ala Ile Ser Ile Ile Thr Ala Leu
145 150 155 160
Thr Glu Phe Thr Glu Phe Asn Gly Ala Thr Arg Val Ile Leu Gly Ser
165 170 175
His Arg Trp Glu Arg Ile Gly Asp Pro Ser Pro Asp Leu Ala Val Arg
180 185 190
Ala Val Met Lys Pro Gly Asp Ala Met Ile Met Tyr Gln Gly Thr Val
195 200 205
His Gly Gly Cys Pro His Asp Glu Gly Asn Pro Glu His Arg Arg Leu
210 215 220
Leu Leu Leu Gly Met Gly Thr Cys Gln Leu Thr Pro Tyr Glu Thr His
225 230 235 240
Met Thr Met Pro Arg Arg Ile Val Glu Ser Met Thr Pro Leu Ala Gln
245 250 255
Lys Met Ile Gly Trp Arg Ser Val Arg Pro Val Ile Ser Asn Val Thr
260 265 270
Gly Leu Met Thr Val Arg Met Lys His Leu Glu Arg Gln Ile Glu Leu
275 280 285
Lys Ser Asp Val Pro Leu Glu Glu Glu Arg
290 295
<210> 101
<211> 300
<212> PRT
<213> Trichophyton rubrum
<400> 101
Met Ser Glu Gln Ile Ile Glu Arg Phe Pro Val Ser Ala Gly Pro Asp
1 5 10 15
Ala Ile Phe Glu Ser Phe Lys Lys Asn Gly Val Val Val Ile Glu Asn
20 25 30
Phe Ala Lys Lys Asp Gln Leu Asp Arg Phe Thr Ala Glu Ile Asp Pro
35 40 45
Ala Leu Gln Lys Leu Thr Ala Gly Tyr Ala Ser Arg Ile Asp Gly Asp
50 55 60
Glu Pro Val Asp Glu Phe Phe Gly Lys Lys Thr Lys Arg Leu Gly Gln
65 70 75 80
Leu Ser Thr Val Ser Ser Val Phe Arg His Glu Phe Leu Glu His Asp
85 90 95
Leu Met His Ser Leu Leu Glu Arg Asn Phe Ile Asp Tyr Pro Leu Ala
100 105 110
Gly Tyr Trp Met Asn Thr Ser Asp Val Ile Glu Ile Gly Pro Gly Ser
115 120 125
Lys Pro Gln Pro Ile His Arg Asp Leu Glu Leu Tyr His Pro Phe Val
130 135 140
Ile Gly Gly Pro Ala Met Pro Glu Ala Ile Cys Asn Phe Met Val Ala
145 150 155 160
Leu Thr Pro Phe Thr Ala Glu Asn Gly Ala Thr Val Phe Ala Pro Gly
165 170 175
Thr His Leu Asn Glu Ser Phe Glu Arg Leu Glu Asp Gly Gly Leu Pro
180 185 190
Gly Gly Asp Lys Thr Ile Ser Ala Val Met Asn Pro Gly Asp Cys Ala
195 200 205
Phe Phe Ser Gly Lys Val Ile His Gly Gly Gly Thr Asn Ser Thr Thr
210 215 220
Asp Glu Phe Arg Arg Gly Leu Ser Met Ser Phe Ile Arg Lys Ile Leu
225 230 235 240
Ser Pro Glu Gln Ala His Pro Leu Ser Ile Ser Arg Glu Ile Val Glu
245 250 255
Thr Met Ser Tyr Arg Gly Gln Ala Met Leu Gly Phe Arg Ser Asn Trp
260 265 270
Pro Ala Ile Gly Asp Glu Pro Ala Ile Ile Trp Ser Tyr Gln Gly Ser
275 280 285
Asp Ile Gly Lys Lys Leu Gly Leu Gly Asp Lys Asn
290 295 300
<210> 102
<211> 1440
<212> DNA
<213> Artificial Sequence
<220>
<223> DmaW of A. japonicus with alternative C-terminus codon optimized
for S. cerrevisiae
<400> 102
atgactgctg gtcaaggtat taagactggt aatgcttctg attgcgaagt ctacagaact 60
ttgtctgttg ctttggattt cgccaatcaa gatgaagaat tgtggtggca ttctactgct 120
ccaatgtttg ctcaaatgtt gcaatctacc aactacaact tacacgctca gtacaagcac 180
ttgttgatct acaagaagaa cgtcattcca ttcttgggtg tttacccaac taatgataag 240
ccaagatggt tgtccatttt gaccagatat ggtactccat tcgaattgtc tttgaactgt 300
tctggtccat tggttagata cacttacgaa cctattaacg ctgctactgg tactgctaga 360
gatccattca atactcatgc tgtttgggac tcattggaac aattgatggc tttacaatcc 420
ggtatcgatt tggatttgtt cagacacttc aagaacgact tgactttgtc tgctgaagaa 480
tccgaatact tgtacaagaa caacttggtc ggtgaacaaa tcagaaccca aaacaaattg 540
gctttggact tgcaagatgg tgaattcgtt gttaagactt acatctaccc agccttgaaa 600
tctttggcta ctggtagaag tatccacgaa ttggtttttg gttctgcttt cagatggtct 660
aagcaatacc cagaattgag aaagccattg gataccttgg aacaatacgt ttattctaga 720
ggtccatctt ctactgcctc tccaagatta ttgtcttgcg atttgattga ccctaccaag 780
tccagaatca aaatctactt gttggaaaga atggtcactt tggaagcctt ggaagatttg 840
tggactatgg gtggtgaaag aactgatgct tctactttgg ctggtttgga aatgattaga 900
gaattgtggg aattgatcag attgccagct ggtttacaat cttatccagc tccatatttg 960
ccaatcggta ctattccaga tgaacaattg ccattgatgg ccaactacac catccatcat 1020
gatgatccag ttccagaacc acaagtttac tttactactt tcggtagaaa cgacatgcaa 1080
attgctgatg ctttggctac tttcttcgaa cgtcgtggtt ggcacgaaat ggctagacag 1140
tacaaagctg aattgtgctc tcattaccca catgctgatc acgaaacttt gaactacttg 1200
cacgcttaca tctcattctc ttacagaaag aacaagccat acttgtccgt ctacttgcaa 1260
tctttggaaa ctggtgattg ggttacctca tccttcaact ctgtccatgt ggaccctggc 1320
ctttcagcca ccgtccaaga gctgtccaag ctgaccaaga ccgccggaac caccgttagg 1380
gagacaaaac taccactaac cccagatgga tcagagcctg gcgtcatcac ccagtactag 1440
<210> 103
<211> 1239
<212> DNA
<213> Artificial Sequence
<220>
<223> EasA of A. japonicus codon optimized for S. cerrevisiae
<400> 103
atgaccatcg atccatcttc tccaggtgct ccagcttcta agttgtttca acctatgaag 60
attggtttgt gccacttgca acatagattg attatggctc caactaccag atacagagct 120
gattctactg ctactccatt gccattcgtt caagaatatt atgctcaaag agctgctgtt 180
ccaggtactt tgttgattac tgaagctacc gatatttctc cacaagctgc tggtgaacca 240
catattccag gtctatattc tgctactcaa attgctgctt ggagaggtat agtttctgct 300
gttcatagaa gaggttgcta cattttctgt caattgtggg ctactggtag agcagctgat 360
cctgctttgt tgaaagaaaa aggtttccca ttggtcagtt cctccgatgt tccaattgaa 420
actgaatctc aacaaccaca accattgacc caagctgaaa ttgaaggtta cattgctgat 480
ttcgttaccg ctgctagagt tgctgttcat gaagttggtt ttgatggtgt tgaattgcat 540
ggtgctaacg gttacttgat tgatcaattc actcaatggc catgcaatca aagaactgat 600
tgctggggtg gttctattga aaacagagct agatttgctt tggctgttac tagagctgtt 660
gttgatgctg ttggtgctga aagagttgga gttaagttgt ctccatggtc acaatatgct 720
ggtatgggtg ttatggctga tttggttcca caattcgaat tcttgattaa gagattgaga 780
gaagagtaca acgacggtcc aggtttggct tacttgcatt tggctaattc tagatggttg 840
gacgaagaaa cttctcatcc agatccacat catgaagttt ttgttagagc ttggagaggt 900
tctggtagag atggtattga agctaaaaaa gaagccccac cagttatttt ggctggtggt 960
tatgatgctg tttctgctcc agaagttgct gacaaggttt tcaaagaaga agataacgtt 1020
gctgttgcct tcggtagata ctttatttct actccagatt tgccattcag aatgcaaaga 1080
ggtattccat tgcaaaagta cgatagagct tctttctaca cctccttatt gaaagaaggt 1140
tacttggact atccatactc cgctgaatat gttgctgaat tcggtcaaag attgaagaag 1200
aaagaagctg ctgctaccgg ttctatttgt caaggttaa 1239
<210> 104
<211> 1530
<212> DNA
<213> Artificial Sequence
<220>
<223> EasC of A. japonicus codon optimized for S. cerrevisiae
<400> 104
atggctccag attctaagcc aacttacact actgctaatg gttgtccatt tcataagcct 60
gaaggtccac cagctgatgg tagaactttg gctttgtctg atcatcattt ggttgatacc 120
ttggcccatt tcaacagaga aaagattcca gaaagagttg ttcacgctaa aggtgctggt 180
gcttatggtg aatttgaagt tactgccgat atctccgata tttgcgatat cgatatgttg 240
gttggtgtcg gtaaaaagac tccatgtgtt actagattct ctaccactgg tttggaaaga 300
ggttctaacg aaggtatgag agatttgaaa ggtatggctt gtaagttcta cactactgaa 360
ggtaattggg attgggtcat gttgaatttc ccattcttct tcattcgtga tccagtcaag 420
ttcccatctt tgatgcacgc tcaaagaaga gatccacaaa ctaatttgtt gaacccaaac 480
atgtactggg attgggttac caacaatcat gaatccttgc acatggtctt gttgcaattt 540
tctgatttcg gtactatgtt caattggaga tccatgtctg gttatatggc tcatgcttac 600
aaatgggtta agccagatgg ttcttttaag tacgtccaca tcttcttgtc atctgataga 660
ggtccaaatt tcaccgatgg tcaacaagct aaaggtacaa atgatttgga tccagatcac 720
gctactagag acttgtatga agctattgaa agaggtgaat acccaacttg gactgcttct 780
gttcaagttg ttgatccaaa agatgctcca aacttgggtt acaacatttt ggatgttact 840
aagcactgga acttgggtac ttacccaaaa gatgttactt tgatcccacc aaaggttttc 900
ggtaagttga ctttgaaaag aaacccagct aactacttcg ccgaaattga acaattggct 960
tactctccat ctaacatggt tccaggtgtt gctccatctg aagatccaat tttacaagct 1020
agaattttcg cttacccaga cgctcaaaga tatagattgg gtgctaacca tcaacaaatc 1080
ccagttaata gatccgctca tacctttaac ccaattgcta gagatggtca aggtactttt 1140
gatgctaatt atggtgctca tccaggtttc ttgactcaac aacaaccagt tagatttgcc 1200
gaacctagag aaccagatcc aaagtataat gaatggttga aagaaatcca atccaagtcc 1260
tggttgcaaa ctactgaaca tgattacaag ttcgccagag atttctacga agttttgcca 1320
gattttagag gtcaagaatt ccaagatacc atggtccaaa acatggttga atctgttgct 1380
caaaccagag ccgaaattca aaagcaagtt tacgaaacct ggaagttggt ttctccagct 1440
ttggctgcta gaattcaaaa aggtgttgaa accttgttgc aaaagtctga agctccagct 1500
gaagaatctt ttattccagc tagattgtga 1530
<210> 105
<211> 789
<212> DNA
<213> Artificial Sequence
<220>
<223> EasD of A. japonicus codon optimized for S. cerrevisiae
<400> 105
atgaccgttg tcaactctag aattttcgct gttactggtg gtgcttctgg tattggtgct 60
gctacttgta gattattggc tgctagaggt gctgctgtta tatgcattgg tgatatttct 120
ccaaagggtt tcgaatccgt tacctctgat attcaagcct tgaatccagc tacaaaggtt 180
cattgcacta gattggatgt ttcttcatcc gctgaagttg atgcttggtt gcaaactatc 240
gttcaaactt ttggtgactt gcatggtttg gctaatgttg ctggtattgc tcaaccagct 300
ggtgctagac aagctccagc tttgttggaa gaaactgatg aagaatggca aagagttttc 360
aaggttaact tggacggtgt tttctactgt actagagctg gtattagagc tatgaaggct 420
ttgccaggtc caaaagatag agctatcgtt aacattggtt ccattgctgc ctttatccat 480
ttgccagatg cttatgctta cggtacttct aaaggtgctt gtgcttactt tacttcttgt 540
gctgctatgg atgcttggcc attgggtatt agaatcaatt ctgtttctcc aggtattacc 600
gatactccag ctttgccaca attcatgcca caagctaaat ctatcgacga agtaaaagaa 660
gcctacatca aagaaggttt cccaatcatt caacctgaag atgttgctag aactatcgtc 720
tggttgttgt ctgaagattc tagaccagtt tacggtgcca atattaacgt tggtgctgca 780
actccataa 789
<210> 106
<211> 1866
<212> DNA
<213> Artificial Sequence
<220>
<223> EasE of A. japonicus codon optimized for S. cerrevisiae
<400> 106
atgggtcaat ccagaggtat tttgggtggt gttagacaat tgatcttggt tattttggtt 60
ggtgcctact tgtctagatt gtctgctgtt gatgatgata gacatgattg cagatgtaga 120
ccaggtgaac catgttggcc aactgtttct cattggtcta ctttgaacga atctattggt 180
ggtgctttgg ctcatgttaa gccaattgct catgtttgtc atcaatccgg tcaagattct 240
tctgcttgcg aacaagtttt acaagaatcc ttggattcta agtggcgtgc ttctcatact 300
ggtgctttac aagattgggt ttgggaaggt ggtgctgaat ctaatcaaac ttgttactac 360
ttgagatcag gtccagctgg ttcttgtcat caaggtagaa ttccattata ttccgctgct 420
gttaagtccg cttctgatgt tcaaaaggtt gttgatttca ccagacaaca caacttgaga 480
ttggttatta gaaacaccgg tcatgatggt tctggtagat catctggtcc agattctgtt 540
gaaattcata cccatcactt gaactccgtt caataccatc caaattttag accagctggt 600
tcctctgaaa gacaatctgc tccaggtcaa ccagctgtta ctgttggtgc tggtattttg 660
ttgggtgact tgtatgctag aggtgcttct gaaggttgga tagttgttgg tggtgaatgt 720
ccaacagttg gtgctgctgg tggtttttta caaggtggtg gtgtttcttc ttggttgtct 780
tatgctcatg gtttggctgt tgataacgtc ttggaatacg aagttgttac tgctaagggt 840
gaaatcgtta ttgctaacgc tcatcaaaac ccagatttgt tttgggcttt gagaggcggt 900
ggtggtggta cttttggtgt tgttactcaa gctacattgc aagttcatcc agatttgcca 960
gtttctgttg ctgatgctgt tgttactggt tctagagctg atgctacttt ttggtcacat 1020
ggtgttgctg ctttgttgag agctttacaa ttcttgaaca accatggtac tgccggtcaa 1080
ttcattttga gaaacactga tgatgatacc gtccaagcta gtttgactat gtacttctct 1140
aacttgactg ttccagctgt tgctgacgaa agaatggatc ctttgagaag agctttggaa 1200
cataatggtc atccatacca attgacctct agattcttgc cacaaatctc ttctactttc 1260
agacataccg ctgatagata cccagaagat tacggtattt tgatgggttc cgttttggtt 1320
tccttggact tgtttaattc tgctactggt ccagctgctt tggcacaaca ttttgctaga 1380
ttgccaatga ctccagatga tttgttgttc acttctaact tgggtggtag agtctcttca 1440
ttgaatagag atccagcttc tactgctatg catccaggtt ggagagatgc tgctcaatta 1500
ttgaatttcg ttagaggtgt tggtgctcca tctttggctg ctaaagctac tgctttacat 1560
gaattgcaaa ccgttcaaat ggccagatta tacgaaattg aaccagcctt ccaaatctcc 1620
tacagaaatt tgggtgatcc atccgaaaga agatccagag aagtttattg gggttctaac 1680
tacgctagat tggttgaagt taagagaaga tgggatccag aaggtttgtt cttctccaaa 1740
ttgggtattg atggtgatgc ttgggatgct gaaggtatgt gtagacaaac aagacaaggt 1800
gcttggaatg ttgctgttaa gtgggttcaa tctttcttgg gttctttgtc tgcttccgtt 1860
gtttga 1866
<210> 107
<211> 1020
<212> DNA
<213> Artificial Sequence
<220>
<223> EasF of A. japonicus codon optimized for S. cerrevisiae Af_optSc
<400> 107
atgacaattt ctgctccacc tattattgat attagacaag cagggttgga aagttctata 60
ccagaccaag ttgtcgaagg tctaactaag gaagtcaaga cacttcctgc acttttgttt 120
tactcaacaa aaggcattca gcattggaat agacattccc acgctgctga tttctatcct 180
agacatgagg aactatgcat cctcaaagct gaagctagta agatggctgc ttcaatcgct 240
caagatagct tagtaatcga tatgggttct gccagtatgg ataaggttat cctcctgttg 300
gaagcgttag aggaacagaa aaagtccata acttactacg ccttggacct gtcctactct 360
gaattagcat ctaattttca agccattcca gtggacagat tccactatgt tagattcgca 420
gccttacatg gcacttttga cgatggactt cactggttac aaaacgctcc tgatattagg 480
aacagaccaa gatgtatact gttatttggg ttaaccatcg gtaacttctc acgagataat 540
gctgcatctt tcctcagaaa tatcgcccaa tctgctctgt ctacatcccc aactcaatca 600
tccattatag tgtctctaga ttcatgtaaa cttcctacga aaatcttaag agcctacacc 660
gcagatggag tagttccatt tgctcttgcc tcactaagct atgcgaattc attgtttcat 720
ccaaaaggcg acagaaagat tttcaatgaa gaggattggt actttcattc tgaatggaac 780
catgcattgg gtcgtcacga agctagtttg atcacacaat caaaagatat ccaattaggt 840
gcaccactag agactgttat agtcagacgt gatgaaaaga tcagatttgg atgctcttac 900
aaatacgaca aagctgaaag ggatcagttg tttcattcag cgggtttgga ggatgccgca 960
gtgtggacag caccagactg tgacgtagca ttctatcaat tgagactaag attgaactaa 1020
<210> 108
<211> 855
<212> DNA
<213> Artificial Sequence
<220>
<223> EasG of A. japonicus codon optimized for S. cerrevisiae
<400> 108
atgaccatct tggttttggg tggtagaggt aaaactgctt ctagattgtc tgctttgttg 60
catgaagctc aaatcccttt tgttgtcggt acttcttcta ctgctccaat tgctacttat 120
agaaccgctc attttgactg gttgaacaag gatacttgga ctaatgcttg ggatcaagct 180
ccagaaccta ttactgctat ctatttggtt ggtgctcatg ctccagaatt tgttcatcca 240
atgttcgaat tcatcgattt ggctcattct agaggtgtta gaagatacgc tttggtttct 300
gcttctaaca ctgaaaaagg tgatccaatg atgggtgaag ctcatgctca tttggattct 360
aaagaaggtg ttaagtacgt cgtcttaaga ccaacttggt ttatggaaaa ccacatcgaa 420
gatccacatt tgtcttggat taagaccgaa aacaaaatct actctgctac tggtccaggt 480
aagattccat ttgtttctgc tgatgatatt gccagagttg cttttcatgc tttgactcaa 540
ccattgaccc aaaaagaaca acaatatatg gttttgggtc cagacttgtt gtcttatgct 600
gatatggctg aaatcttgac cgatttgttg ggtagaaaga ttacccatgt tactttgcca 660
gctgaaaagt tcggtcaatt cttggttgat actgctggtt tgccaccaga ttttgctgaa 720
atgttggcta ctatggaagc tgctgttgct actggtgctg aagaaagagt ttctcaagac 780
gttgaaaagg ttactggtag accacctaga agatttgttg attacgctag agaaaacaga 840
gaacattggt tgtaa 855
<210> 109
<211> 882
<212> DNA
<213> Artificial Sequence
<220>
<223> EasH of A. japonicus codon optimized for S. cerrevisiae
<400> 109
atgaccatta ccgaaacttc taagccaacc ttgagaagaa ttccaagatc agctggtgat 60
gaagctattt tccaagtctt gcaagaagat ggcgttgttg ttatcgaagg tttcatgtct 120
gctgaccaag ttagaagatt caacggtgaa attgatccac acatgaagca atgggaattg 180
ggtcaaaaat cctaccaaga atcttacttg gctggtatga gacaattgtc atctttgcct 240
ttgttctcca agttgttcag agatgaattg atgaacgacg aattattgca cggtttgtgc 300
aagagattat tcggtccaga atctggtgat tactggttga ctacttcttc tgttttggaa 360
actgaaccag gttaccatgg tcaagaattg catagagaac atgatggtat tccaatctgt 420
actaccttgg gtagacaatc tccagaatct atgttgaact tcttgactgc tttgactgat 480
ttcactgctg aaaatggtgc tactagagtt ttgccaggtt ctcatttgtg ggaagatttt 540
tctgctccac caccaaaagc tgatactgct attccagctg ttatgaatcc aggtgatgct 600
gttttgttta ctggtaaaac tttacatggt gccggtaaga acaacactac tgattttttg 660
agaagaggtt tcccattgat catgcaatct tgtcaattca ctccagttga agctagtgtt 720
gctttgccaa gagaattggt tgaaactatg actccattgg cccaaaaaat ggttggttgg 780
agaactgttt ctgctaaggg tgttgatatt tggacctacg atttgaagga tttggctacc 840
ggtattgact tgaagtctaa tcaagttgct aagaaggcct aa 882
<210> 110
<211> 1801
<212> DNA
<213> Artificial Sequence
<220>
<223> P/A cassette including both Asc I sites
<400> 110
ggcgcgccat gcagggatat cagatctacg cacagatatt ataacatctg cataataggc 60
atttgcaaga attactcgtg agtaaggaaa gagtgaggaa ctatcgcata cctgcattta 120
aagatgccga tttgggcgcg aatcctttat tttggcttca ccctcatact attatcaggg 180
ccagaaaaag gaagtgtttc cctccttctt gaattgatgt taccctcata aagcacgtgg 240
cctcttatcg agaaagaaat taccgtcgct cgtgatttgt ttgcaaaaag aacaaaactg 300
aaaaaaccca gacacgctcg acttcctgtc ttcctattga ttgcagcttc caatttcgtc 360
acacaacaag gtcctagcga cggctcacag gttttgtaac aagcaatcga aggttctgga 420
atggcgggaa agggtttagt accacatgct atgatgccca ctgtgatctc cagagcaaag 480
ttcgttcgat cgtactgtta ctctctctct ttcaaacaga attgtccgaa tcgtgtgaca 540
acaacagcct gttctcacac actcttttct tctaaccaag ggggtggttt agtttagtag 600
aacctcgtga aacttacatt tacatatata taaacttgca taaattggtc aatgcaagaa 660
atacatattt ggtcttttct aattcgtagt ttttcaagtt cttagatgct ttctttttct 720
cttttttaca gatcatcaag gaagtaatta tctacttttt acaacaaata taaaacaaag 780
cttggcctgc agggccagct tacccttaaa tttatttgca ctactggaaa actacctgtt 840
ccatggccaa cacttgtcac tactttctct tatggtgttc aatgcttttc ccgttatccg 900
gatcatatga aacggcatga ctttttcaag agtgccatgc ccgaaggtta tgtacaggaa 960
cgcactatat ctttcaaaga tgacgggaac tacaagacgc gtgctgaagt caagtttgaa 1020
ggtgataccc ttgttaatcg tatcgagtta aaaggtattg attttaaaga agatggaaac 1080
attctcggac acaaactcga gtacaactat aactcacaca atgtatacat cacggcagac 1140
aaacaaaaga atggaatcaa agctaacttc aaaattcgcc acaacattga agatggatcc 1200
gttcaactag cagaccatta tcaacaaaat actccaattg gcgatggccc tgtcctttta 1260
ccagacaacc attacctgtc gacacaatct gccctttcga aagatcccaa cgaaaagcgt 1320
gaccacatgg tccttcttga gtttgtaact gctgctggga ttacacatgg catggatgag 1380
ctctacaaat aatgaattcg tcgaggccgt tcaggcctcg aggccgttca ggctcgaccc 1440
ggggatccgc ggatctctta tgtctttacg atttatagtt ttcattatca agtatgccta 1500
tattagtata tagcatcttt agatgacagt gttcgaagtt tcacgaataa aagataatat 1560
tctacttttt gctcccaccg cgtttgctag cacgagtgaa caccatccct cgcctgtgag 1620
ttgtacccat tcctctaaac tgtagacatg gtagcttcag cagtgttcgt tatgtacggc 1680
atcctccaac aaacagtcgg ttatagtttg tcctgctcct ctgaatcgtc tccctcgata 1740
tttctcattt tccttcgcat gcccatgggt taactgatca atgcatcctg catggcgcgc 1800
c 1801
<210> 111
<211> 1746
<212> DNA
<213> Artificial Sequence
<220>
<223> G/C cassette including both AscI sites
<400> 111
ggcgcgccat gcagggatat cagatctcag ttcgagttta tcattatcaa tactgccatt 60
tcaaagaata cgtaaataat taatagtagt gattttccta actttattta gtcaaaaaat 120
tagcctttta attctgctgt aacccgtaca tgcccaaaat agggggcggg ttacacagaa 180
tatataacat cgtaggtgtc tgggtgaaca gtttattcct ggcatccact aaatataatg 240
gagcccgctt tttaagctgg catccagaaa aaaaaagaat cccagcacca aaatattgtt 300
ttcttcacca accatcagtt cataggtcca ttctcttagc gcaactacag agaacagggg 360
cacaaacagg caaaaaacgg acacaacctc aatggagtga tgcaacctgc ctggagtaaa 420
tgatgacaca aggcaattga cccacgcatg tatctatctc attttcttac accttctatt 480
accttctgct ctctctgatt tggaaaaagc tgaaaaaaaa ggttgaaacc agttccctga 540
aattattccc ctacttgact aataagtata taaagacggt aggtattgat tgtaattctg 600
taaatctatt tcttaaactt cttaaattct acttttatag ttagtctttt ttttagtttt 660
aaaacaccaa gaacttagtt tcgaataaac acacataaac aaacaaaaag cttggcctgc 720
agggccagct tacccttaaa tttatttgca ctactggaaa actacctgtt ccatggccaa 780
cacttgtcac tactttctct tatggtgttc aatgcttttc ccgttatccg gatcatatga 840
aacggcatga ctttttcaag agtgccatgc ccgaaggtta tgtacaggaa cgcactatat 900
ctttcaaaga tgacgggaac tacaagacgc gtgctgaagt caagtttgaa ggtgataccc 960
ttgttaatcg tatcgagtta aaaggtattg attttaaaga agatggaaac attctcggac 1020
acaaactcga gtacaactat aactcacaca atgtatacat cacggcagac aaacaaaaga 1080
atggaatcaa agctaacttc aaaattcgcc acaacattga agatggatcc gttcaactag 1140
cagaccatta tcaacaaaat actccaattg gcgatggccc tgtcctttta ccagacaacc 1200
attacctgtc gacacaatct gccctttcga aagatcccaa cgaaaagcgt gaccacatgg 1260
tccttcttga gtttgtaact gctgctggga ttacacatgg catggatgag ctctacaaat 1320
aatgaattcg tcgaggccgt tcaggcctcg aggccgttca ggctcgaccc ggggatccgc 1380
ggcacgtccg acggcggccc gacgggtccg aggcctcgga gatccgtccc ccttttcctt 1440
tgtcgatatc atgtaattag ttatgtcacg cttacattca cgccctcccc ccacatccgc 1500
tctaaccgaa aaggaaggag ttagacaacc tgaagtctag gtccctattt atttttttat 1560
agttatgtta gtattaagaa cgttatttat atttcaaatt tttctttttt ttctgtacag 1620
acgcgtgtac gcatgtaaca ttatactgaa aaccttgctt gagaaggttt tgggacgctc 1680
gaaggcttta atttgcaagc tgcatgccca tgggttaact gatcaatgca tcctgcatgg 1740
cgcgcc 1746
<210> 112
<211> 2432
<212> DNA
<213> Artificial Sequence
<220>
<223> pUC18 into which an Asc I site (SeqID 113) was inserted into the
Pvu II sites
<400> 112
tcgcgcgttt cggtgatgac ggtgaaaacc tctgacacat gcagctcccg gagacggtca 60
cagcttgtct gtaagcggat gccgggagca gacaagcccg tcagggcgcg tcagcgggtg 120
ttggcgggtg tcggggctgg cttaactatg cggcatcaga gcagattgta ctgagagtgc 180
accatatgcg gtgtgaaata ccgcacagat gcgtaaggag aaaataccgc atcaggcgcc 240
attcgccatt caggctgcgc aactgttggg aagggcgatc ggtgcgggcc tcttcgctat 300
tacgccagct gatttgcccg ggcagttcag gctcatcagg cgcgcctgat gagcctgaac 360
tgcccgggca aatcagctgc attaatgaat cggccaacgc gcggggagag gcggtttgcg 420
tattgggcgc tcttccgctt cctcgctcac tgactcgctg cgctcggtcg ttcggctgcg 480
gcgagcggta tcagctcact caaaggcggt aatacggtta tccacagaat caggggataa 540
cgcaggaaag aacatgtgag caaaaggcca gcaaaaggcc aggaaccgta aaaaggccgc 600
gttgctggcg tttttccata ggctccgccc ccctgacgag catcacaaaa atcgacgctc 660
aagtcagagg tggcgaaacc cgacaggact ataaagatac caggcgtttc cccctggaag 720
ctccctcgtg cgctctcctg ttccgaccct gccgcttacc ggatacctgt ccgcctttct 780
cccttcggga agcgtggcgc tttctcatag ctcacgctgt aggtatctca gttcggtgta 840
ggtcgttcgc tccaagctgg gctgtgtgca cgaacccccc gttcagcccg accgctgcgc 900
cttatccggt aactatcgtc ttgagtccaa cccggtaaga cacgacttat cgccactggc 960
agcagccact ggtaacagga ttagcagagc gaggtatgta ggcggtgcta cagagttctt 1020
gaagtggtgg cctaactacg gctacactag aaggacagta tttggtatct gcgctctgct 1080
gaagccagtt accttcggaa aaagagttgg tagctcttga tccggcaaac aaaccaccgc 1140
tggtagcggt ggtttttttg tttgcaagca gcagattacg cgcagaaaaa aaggatctca 1200
agaagatcct ttgatctttt ctacggggtc tgacgctcag tggaacgaaa actcacgtta 1260
agggattttg gtcatgagat tatcaaaaag gatcttcacc tagatccttt taaattaaaa 1320
atgaagtttt aaatcaatct aaagtatata tgagtaaact tggtctgaca gttaccaatg 1380
cttaatcagt gaggcaccta tctcagcgat ctgtctattt cgttcatcca tagttgcctg 1440
actccccgtc gtgtagataa ctacgatacg ggagggctta ccatctggcc ccagtgctgc 1500
aatgataccg cgagacccac gctcaccggc tccagattta tcagcaataa accagccagc 1560
cggaagggcc gagcgcagaa gtggtcctgc aactttatcc gcctccatcc agtctattaa 1620
ttgttgccgg gaagctagag taagtagttc gccagttaat agtttgcgca acgttgttgc 1680
cattgctaca ggcatcgtgg tgtcacgctc gtcgtttggt atggcttcat tcagctccgg 1740
ttcccaacga tcaaggcgag ttacatgatc ccccatgttg tgcaaaaaag cggttagctc 1800
cttcggtcct ccgatcgttg tcagaagtaa gttggccgca gtgttatcac tcatggttat 1860
ggcagcactg cataattctc ttactgtcat gccatccgta agatgctttt ctgtgactgg 1920
tgagtactca accaagtcat tctgagaata gtgtatgcgg cgaccgagtt gctcttgccc 1980
ggcgtcaata cgggataata ccgcgccaca tagcagaact ttaaaagtgc tcatcattgg 2040
aaaacgttct tcggggcgaa aactctcaag gatcttaccg ctgttgagat ccagttcgat 2100
gtaacccact cgtgcaccca actgatcttc agcatctttt actttcacca gcgtttctgg 2160
gtgagcaaaa acaggaaggc aaaatgccgc aaaaaaggga ataagggcga cacggaaatg 2220
ttgaatactc atactcttcc tttttcaata ttattgaagc atttatcagg gttattgtct 2280
catgagcgga tacatatttg aatgtattta gaaaaataaa caaatagggg ttccgcgcac 2340
atttccccga aaagtgccac ctgacgtcta agaaaccatt attatcatga cattaaccta 2400
taaaaatagg cgtatcacga ggccctttcg tc 2432
<210> 113
<211> 74
<212> DNA
<213> Artificial Sequence
<220>
<223> Linker inserted into Pvu II sites of pUC18, containing an Asc I
site
<400> 113
cagctgattt gcccgggcag ttcaggctca tcaggcgcgc ctgatgagcc tgaactgccc 60
gggcaaatca gctg 74
<210> 114
<211> 10062
<212> DNA
<213> Artificial Sequence
<220>
<223> pEVE2294 (YORW?17-KanMX-G/C+Aj_DmaW_altC-P/A+Af_EasF)
<400> 114
cggtgcgggc ctcttcgcta ttacgccagc tggcgaaagg gggatgtgct gcaaggcgat 60
taagttgggt aacgccaggg ttttcccagt cacgacgttg taaaacgacg gccagtgaat 120
tcgagctccc tgcaggtgtg cacaaaggcc ataatattat gtctacagaa tatactagat 180
gtcctcccta taggatatag taatcctcta aatggaaccg atatttctac ataataatat 240
tacgattatt cctccttccg ttttatatgt ttcattatcc tagcacacta tcaatctttg 300
catttcagct tccattagat ttgatgacta tttctcaatc tttatgttat ctccttacgc 360
cgcatgtgat aatatactgc tagtatgact actagttgat agaagatagt tgatttttac 420
tccaacaaaa gtaacaatat tatttagaac tatagattcc attttgtgca ttcccatatt 480
ctcgaggaaa acttttagta tattctgtag acataatatt atcgcctttg tgaacaatag 540
aatcccaaca attgtcgcaa atttaccaat tttctagatt gcagtcacct tttcaattaa 600
tcactagtgt ttcacttgta acattgtcgt tgttgtttaa cgtattctgt cccgtgccaa 660
ctatgacaaa aatgcaatga tttcagcggt taaatacgaa gcgcaacaag agttagcgaa 720
aaataagtac caccattcta cgctaccatt acttactgaa attagagaca actgttatct 780
attggcagat gttcatacgg ggctttcaaa tattgatgaa attatgtgat gtttagaaga 840
agattcgaac tgttttcagt agatttggta actgtgcaac cataactcat gccgcggccg 900
cataggccac tagtggatct gatatcacct aataacttcg tatagcatac attatacgaa 960
gttatattaa gggttctcga gagctcgttt tcgacactgg atggcggcgt tagtatcgaa 1020
tcgacagcag tatagcgacc agcattcaca tacgattgac gcatgatatt actttctgcg 1080
cacttaactt cgcatctggg cagatgatgt cgaggcgaaa aaaaatataa atcacgctaa 1140
catttgatta aaatagaaca actacaatat aaaaaaacta tacaaatgac aagttcttga 1200
aaacaagaat ctttttattg tcagtactga ttagaaaaac tcatcgagca tcaaatgaaa 1260
ctgcaattta ttcatatcag gattatcaat accatatttt tgaaaaagcc gtttctgtaa 1320
tgaaggagaa aactcaccga ggcagttcca taggatggca agatcctggt atcggtctgc 1380
gattccgact cgtccaacat caatacaacc tattaatttc ccctcgtcaa aaataaggtt 1440
atcaagtgag aaatcaccat gagtgacgac tgaatccggt gagaatggca aaagcttatg 1500
catttctttc cagacttgtt caacaggcca gccattacgc tcgtcatcaa aatcactcgc 1560
atcaaccaaa ccgttattca ttcgtgattg cgcctgagcg agacgaaata cgcgatcgct 1620
gttaaaagga caattacaaa caggaatcga atgcaaccgg cgcaggaaca ctgccagcgc 1680
atcaacaata ttttcacctg aatcaggata ttcttctaat acctggaatg ctgttttgcc 1740
ggggatcgca gtggtgagta accatgcatc atcaggagta cggataaaat gcttgatggt 1800
cggaagaggc ataaattccg tcagccagtt tagtctgacc atctcatctg taacatcatt 1860
ggcaacgcta cctttgccat gtttcagaaa caactctggc gcatcgggct tcccatacaa 1920
tcgatagatt gtcgcacctg attgcccgac attatcgcga gcccatttat acccatataa 1980
atcagcatcc atgttggaat ttaatcgcgg cctcgaaacg tgagtctttt ccttacccat 2040
ggttgtttat gttcggatgt gatgtgagaa ctgtatccta gcaagatttt aaaaggaagt 2100
atatgaaaga agaacctcag tggcaaatcc taacctttta tatttctcta caggggcgcg 2160
gcgtggggac aattcaacgc gtctgtgagg ggagcgtttc cctgctcgca ggtctgcagc 2220
gaggagccgt aatttttgct tcgcgccgtg cggccatcaa aatgtatgga tgcaaatgat 2280
tatacatggg gatgtatggg ctaaatgtac gggcgacagt cacatcatgc ccctgagctg 2340
cgcacgtcaa gactgtcaag gagggtattc tgggcctcca tgtcgctggc cgggtgaccc 2400
ggcggggacg aggcaagcta aacagatctc tagacctaat aacttcgtat agcatacatt 2460
atacgaagtt atattaaggg ttgtcgacct gcagcgtacg aagcttcagc tggcggccgc 2520
ggcgcgccag cttgcaaatt aaagccttcg agcgtcccaa aaccttctca agcaaggttt 2580
tcagtataat gttacatgcg tacacgcgtc tgtacagaaa aaaaagaaaa atttgaaata 2640
taaataacgt tcttaatact aacataacta taaaaaaata aatagggacc tagacttcag 2700
gttgtctaac tccttccttt tcggttagag cggatgtggg gggagggcgt gaatgtaagc 2760
gtgacataac taattacatg atatcgacaa aggaaaaggg ggacggatct ccgaggcctc 2820
ggacccgtcg ggccgccgtc ggacgtgccg cggctagtac tgggtgatga cgccaggctc 2880
tgatccatct ggggttagtg gtagttttgt ctccctaacg gtggttccgg cggtcttggt 2940
cagcttggac agctcttgga cggtggctga aaggccaggg tccacatgga cagagttgaa 3000
ggatgaggta acccaatcac cagtttccaa agattgcaag tagacggaca agtatggctt 3060
gttctttctg taagagaatg agatgtaagc gtgcaagtag ttcaaagttt cgtgatcagc 3120
atgtgggtaa tgagagcaca attcagcttt gtactgtcta gccatttcgt gccaaccacg 3180
acgttcgaag aaagtagcca aagcatcagc aatttgcatg tcgtttctac cgaaagtagt 3240
aaagtaaact tgtggttctg gaactggatc atcatgatgg atggtgtagt tggccatcaa 3300
tggcaattgt tcatctggaa tagtaccgat tggcaaatat ggagctggat aagattgtaa 3360
accagctggc aatctgatca attcccacaa ttctctaatc atttccaaac cagccaaagt 3420
agaagcatca gttctttcac cacccatagt ccacaaatct tccaaggctt ccaaagtgac 3480
cattctttcc aacaagtaga ttttgattct ggacttggta gggtcaatca aatcgcaaga 3540
caataatctt ggagaggcag tagaagatgg acctctagaa taaacgtatt gttccaaggt 3600
atccaatggc tttctcaatt ctgggtattg cttagaccat ctgaaagcag aaccaaaaac 3660
caattcgtgg atacttctac cagtagccaa agatttcaag gctgggtaga tgtaagtctt 3720
aacaacgaat tcaccatctt gcaagtccaa agccaatttg ttttgggttc tgatttgttc 3780
accgaccaag ttgttcttgt acaagtattc ggattcttca gcagacaaag tcaagtcgtt 3840
cttgaagtgt ctgaacaaat ccaaatcgat accggattgt aaagccatca attgttccaa 3900
tgagtcccaa acagcatgag tattgaatgg atctctagca gtaccagtag cagcgttaat 3960
aggttcgtaa gtgtatctaa ccaatggacc agaacagttc aaagacaatt cgaatggagt 4020
accatatctg gtcaaaatgg acaaccatct tggcttatca ttagttgggt aaacacccaa 4080
gaatggaatg acgttcttct tgtagatcaa caagtgcttg tactgagcgt gtaagttgta 4140
gttggtagat tgcaacattt gagcaaacat tggagcagta gaatgccacc acaattcttc 4200
atcttgattg gcgaaatcca aagcaacaga caaagttctg tagacttcgc aatcagaagc 4260
attaccagtc ttaatacctt gaccagcagt cattttaagc tttttgtttg tttatgtgtg 4320
tttattcgaa actaagttct tggtgtttta aaactaaaaa aaagactaac tataaaagta 4380
gaatttaaga agtttaagaa atagatttac agaattacaa tcaataccta ccgtctttat 4440
atacttatta gtcaagtagg ggaataattt cagggaactg gtttcaacct tttttttcag 4500
ctttttccaa atcagagaga gcagaaggta atagaaggtg taagaaaatg agatagatac 4560
atgcgtgggt caattgcctt gtgtcatcat ttactccagg caggttgcat cactccattg 4620
aggttgtgtc cgttttttgc ctgtttgtgc ccctgttctc tgtagttgcg ctaagagaat 4680
ggacctatga actgatggtt ggtgaagaaa acaatatttt ggtgctggga ttcttttttt 4740
ttctggatgc cagcttaaaa agcgggctcc attatattta gtggatgcca ggaataaact 4800
gttcacccag acacctacga tgttatatat tctgtgtaac ccgcccccta ttttgggcat 4860
gtacgggtta cagcagaatt aaaaggctaa ttttttgact aaataaagtt aggaaaatca 4920
ctactattaa ttatttacgt attctttgaa atggcagtat tgataatgat aaactcgaac 4980
tgacgcacag atattataac atctgcataa taggcatttg caagaattac tcgtgagtaa 5040
ggaaagagtg aggaactatc gcatacctgc atttaaagat gccgatttgg gcgcgaatcc 5100
tttattttgg cttcaccctc atactattat cagggccaga aaaaggaagt gtttccctcc 5160
ttcttgaatt gatgttaccc tcataaagca cgtggcctct tatcgagaaa gaaattaccg 5220
tcgctcgtga tttgtttgca aaaagaacaa aactgaaaaa acccagacac gctcgacttc 5280
ctgtcttcct attgattgca gcttccaatt tcgtcacaca acaaggtcct agcgacggct 5340
cacaggtttt gtaacaagca atcgaaggtt ctggaatggc gggaaagggt ttagtaccac 5400
atgctatgat gcccactgtg atctccagag caaagttcgt tcgatcgtac tgttactctc 5460
tctctttcaa acagaattgt ccgaatcgtg tgacaacaac agcctgttct cacacactct 5520
tttcttctaa ccaagggggt ggtttagttt agtagaacct cgtgaaactt acatttacat 5580
atatataaac ttgcataaat tggtcaatgc aagaaataca tatttggtct tttctaattc 5640
gtagtttttc aagttcttag atgctttctt tttctctttt ttacagatca tcaaggaagt 5700
aattatctac tttttacaac aaatataaaa caaagcttaa aatgacaatt tctgctccac 5760
ctattattga tattagacaa gcagggttgg aaagttctat accagaccaa gttgtcgaag 5820
gtctaactaa ggaagtcaag acacttcctg cacttttgtt ttactcaaca aaaggcattc 5880
agcattggaa tagacattcc cacgctgctg atttctatcc tagacatgag gaactatgca 5940
tcctcaaagc tgaagctagt aagatggctg cttcaatcgc tcaagatagc ttagtaatcg 6000
atatgggttc tgccagtatg gataaggtta tcctcctgtt ggaagcgtta gaggaacaga 6060
aaaagtccat aacttactac gccttggacc tgtcctactc tgaattagca tctaattttc 6120
aagccattcc agtggacaga ttccactatg ttagattcgc agccttacat ggcacttttg 6180
acgatggact tcactggtta caaaacgctc ctgatattag gaacagacca agatgtatac 6240
tgttatttgg gttaaccatc ggtaacttct cacgagataa tgctgcatct ttcctcagaa 6300
atatcgccca atctgctctg tctacatccc caactcaatc atccattata gtgtctctag 6360
attcatgtaa acttcctacg aaaatcttaa gagcctacac cgcagatgga gtagttccat 6420
ttgctcttgc ctcactaagc tatgcgaatt cattgtttca tccaaaaggc gacagaaaga 6480
ttttcaatga agaggattgg tactttcatt ctgaatggaa ccatgcattg ggtcgtcacg 6540
aagctagttt gatcacacaa tcaaaagata tccaattagg tgcaccacta gagactgtta 6600
tagtcagacg tgatgaaaag atcagatttg gatgctctta caaatacgac aaagctgaaa 6660
gggatcagtt gtttcattca gcgggtttgg aggatgccgc agtgtggaca gcaccagact 6720
gtgacgtagc attctatcaa ttgagactaa gattgaacta accgcggatc tcttatgtct 6780
ttacgattta tagttttcat tatcaagtat gcctatatta gtatatagca tctttagatg 6840
acagtgttcg aagtttcacg aataaaagat aatattctac tttttgctcc caccgcgttt 6900
gctagcacga gtgaacacca tccctcgcct gtgagttgta cccattcctc taaactgtag 6960
acatggtagc ttcagcagtg ttcgttatgt acggcatcct ccaacaaaca gtcggttata 7020
gtttgtcctg ctcctctgaa tcgtctccct cgatatttct cattttcctt cacgcgccaa 7080
tcgtccccaa caaaagtggg ctctcaaaat tcatcacatt taaatgcata taggaagagc 7140
aacagttggt ttgcatctga tgttccttaa agatttcgac ataatgtgcg aagtagataa 7200
aatgggtcat ttattaatag ttatttcatt attaaccagt tgtggtacaa atgcaactaa 7260
agaaaaaaac tactaaacta tccgggaaat gcgccttaga ttgcacttct taattcttat 7320
tttcgatttt tatttttcct ttgataatca taaagagaaa cgacgatcat ttctaaagcc 7380
atttctgcta gtataccgtt aaataagaaa aataaagcca aatattataa tttttctaat 7440
gtgaatccat aaatatcaaa gcatgcaaaa agggaaagaa gtaatgtctt ggatttatat 7500
agcgtatttg tctaagggga gccagctttc ctgcagggca tgcaagcttg gcgtaatcat 7560
ggtcatagct gtttcctgtg tgaaattgtt atccgctcac aattccacac aacatacgag 7620
ccggaagcat aaagtgtaaa gcctggggtg cctaatgagt gagctaactc acattaattg 7680
cgttgcgctc actgcccgct ttccagtcgg gaaacctgtc gtgccagctg cattaatgaa 7740
tcggccaacg cgcggggaga ggcggtttgc gtattgggcg ctcttccgct tcctcgctca 7800
ctgactcgct gcgctcggtc gttcggctgc ggcgagcggt atcagctcac tcaaaggcgg 7860
taatacggtt atccacagaa tcaggggata acgcaggaaa gaacatgtga gcaaaaggcc 7920
agcaaaaggc caggaaccgt aaaaaggccg cgttgctggc gtttttccat aggctccgcc 7980
cccctgacga gcatcacaaa aatcgacgct caagtcagag gtggcgaaac ccgacaggac 8040
tataaagata ccaggcgttt ccccctggaa gctccctcgt gcgctctcct gttccgaccc 8100
tgccgcttac cggatacctg tccgcctttc tcccttcggg aagcgtggcg ctttctcata 8160
gctcacgctg taggtatctc agttcggtgt aggtcgttcg ctccaagctg ggctgtgtgc 8220
acgaaccccc cgttcagccc gaccgctgcg ccttatccgg taactatcgt cttgagtcca 8280
acccggtaag acacgactta tcgccactgg cagcagccac tggtaacagg attagcagag 8340
cgaggtatgt aggcggtgct acagagttct tgaagtggtg gcctaactac ggctacacta 8400
gaaggacagt atttggtatc tgcgctctgc tgaagccagt taccttcgga aaaagagttg 8460
gtagctcttg atccggcaaa caaaccaccg ctggtagcgg tggttttttt gtttgcaagc 8520
agcagattac gcgcagaaaa aaaggatctc aagaagatcc tttgatcttt tctacggggt 8580
ctgacgctca gtggaacgaa aactcacgtt aagggatttt ggtcatgaga ttatcaaaaa 8640
ggatcttcac ctagatcctt ttaaattaaa aatgaagttt taaatcaatc taaagtatat 8700
atgagtaaac ttggtctgac agttaccaat gcttaatcag tgaggcacct atctcagcga 8760
tctgtctatt tcgttcatcc atagttgcct gactccccgt cgtgtagata actacgatac 8820
gggagggctt accatctggc cccagtgctg caatgatacc gcgagaccca cgctcaccgg 8880
ctccagattt atcagcaata aaccagccag ccggaagggc cgagcgcaga agtggtcctg 8940
caactttatc cgcctccatc cagtctatta attgttgccg ggaagctaga gtaagtagtt 9000
cgccagttaa tagtttgcgc aacgttgttg ccattgctac aggcatcgtg gtgtcacgct 9060
cgtcgtttgg tatggcttca ttcagctccg gttcccaacg atcaaggcga gttacatgat 9120
cccccatgtt gtgcaaaaaa gcggttagct ccttcggtcc tccgatcgtt gtcagaagta 9180
agttggccgc agtgttatca ctcatggtta tggcagcact gcataattct cttactgtca 9240
tgccatccgt aagatgcttt tctgtgactg gtgagtactc aaccaagtca ttctgagaat 9300
agtgtatgcg gcgaccgagt tgctcttgcc cggcgtcaat acgggataat accgcgccac 9360
atagcagaac tttaaaagtg ctcatcattg gaaaacgttc ttcggggcga aaactctcaa 9420
ggatcttacc gctgttgaga tccagttcga tgtaacccac tcgtgcaccc aactgatctt 9480
cagcatcttt tactttcacc agcgtttctg ggtgagcaaa aacaggaagg caaaatgccg 9540
caaaaaaggg aataagggcg acacggaaat gttgaatact catactcttc ctttttcaat 9600
attattgaag catttatcag ggttattgtc tcatgagcgg atacatattt gaatgtattt 9660
agaaaaataa acaaataggg gttccgcgca catttccccg aaaagtgcca cctgacgtct 9720
aagaaaccat tattatcatg acattaacct ataaaaatag gcgtatcacg aggccctttc 9780
gtctcgcgcg tttcggtgat gacggtgaaa acctctgaca catgcagctc ccggagacgg 9840
tcacagcttg tctgtaagcg gatgccggga gcagacaagc ccgtcagggc gcgtcagcgg 9900
gtgttggcgg gtgtcggggc tggcttaact atgcggcatc agagcagatt gtactgagag 9960
tgcaccatat gcggtgtgaa ataccgcaca gatgcgtaag gagaaaatac cgcatcaggc 10020
gccattcgcc attcaggctg cgcaactgtt gggaagggcg at 10062
<210> 115
<211> 11142
<212> DNA
<213> Artificial Sequence
<220>
<223> pEVE2312 (YPRC?15-HygR-P/A+Aj_EasC-G/C+Aj_EasE)
<400> 115
cggtgcgggc ctcttcgcta ttacgccagc tggcgaaagg gggatgtgct gcaaggcgat 60
taagttgggt aacgccaggg ttttcccagt cacgacgttg taaaacgacg gccagtgaat 120
tcgagctccc tgcagggcca ggcgccttta tatcatataa ttaagacaca aaaggataaa 180
acaaaggtgt taactattct gcatactcac tatcgtaaac tgtcctgcaa atcgtgtaaa 240
tatgtatttc attttttttg cagtgaaaaa aggcatgtaa aataccgcat caagtaactc 300
tactccgcct gtggtttcaa gactaacggc ttgagacaaa atgggaagaa atgattgcag 360
aaaagccata tgtgtaatag caaaaagctg gatagtgctt accagatgtt taccttaatt 420
tcttggtgaa ttagagaagt acagaagttt tactattaat cccaccatag aaatttgtat 480
aggaaagtag tttattggag ttattggata tactgtgtaa actatttctt gaaattgtaa 540
tcttaagatg ctcttcttat tctattaaaa atagaaaatg attttcatat ttatttattt 600
atttatattt tggcattact cttcatcatt tttttccctc taagaagctt cctttctttt 660
tataaggata acaaaaccaa aaggaatatt gggtcagatg aatggacgcg aatgcaagac 720
agaagtccaa atcacgtcaa gacaaagaaa gaaagaaaga aaaactaaca cattaatgta 780
gttttaaaat ttcaaatccg aacaacagag catagggttt cgcaaagcgg ccgctaactt 840
cgtataatgt atgctatacg aagttatttc gacactggat ggcggcgtta gtatcgaatc 900
gacagcagta tagcgaccag cattcacata cgattgacgc atgatattac tttctgcgca 960
cttaacttcg catctgggca gatgatgtcg aggcaaaaaa aatataaatc acgctaacat 1020
ttgattaaaa tagaacaact acaatataaa aaaactatac aaatgacaag ttcttgaaaa 1080
caagaatctt tttattgtca gtactgatta ttcctttgcc ctcggacgag tgctggggcg 1140
tcggtttcca ctatcggcga gtacttctac acagccatcg gtccagacgg ccgcgcttct 1200
gcgggcgatt tgtgtacgcc cgacagtccc ggctccggat cggacgattg cgtcgcatcg 1260
accctgcgcc caagctgcat catcgaaatt gccgtcaacc aagctctgat agagttggtc 1320
aagaccaatg cggagcatat acgcccggag ccgcggcgat cctgcaagct ccggatgcct 1380
ccgctcgaag tagcgcgtct gctgctccat acaagccaac cacggcctcc agaagaagat 1440
gttggcgacc tcgtattggg aatccccgaa catcgcctcg ctccagtcaa tgaccgctgt 1500
tatgcggcca ttgtccgtca ggacattgtt ggagccgaaa tccgcgtgca cgaggtgccg 1560
gacttcgggg cagtcctcgg cccaaagcat cagctcatcg agagcctgcg cgacggacgc 1620
actgacggtg tcgtccatca cagtttgcca gtgatacaca tggggatcag caatcgcgca 1680
tatgaaatca cgccatgtag tgtattgacc gattccttgc ggtccgaatg ggccgaaccc 1740
gctcgtctgg ctaagatcgg ccgcagcgat cgcatccatg gcctccgcga ccggctgcag 1800
aacagcgggc agttcggttt caggcaggtc ttgcaacgtg acaccctgtg cacggcggga 1860
gatgcaatag gtcaggctct cgctgaattc cccaatgtca agcacttccg gaatcgggag 1920
cgcggccgat gcaaagtgcc gataaacata acgatctttg tagaaaccat cggcgcagct 1980
atttacccgc aggacatatc cacgccctcc tacatcgaag ctgaaagcac gagattcttc 2040
gccctccgag agctgcatca ggtcggagac gctgtcgaac ttttcgatca gaaacttctc 2100
gacagacgtc gcggtgagtt caggcttttt acccatggtt gtttatgttc ggatgtgatg 2160
tgagaactgt atcctagcaa gattttaaaa ggaagtatat gaaagaagaa cctcagtggc 2220
aaatcctaac cttttatatt tctctacagg ggcgcggcgt ggggacaatt caacgcgtct 2280
gtgaggggag cgtttccctg ctcgcaggtc tgcagcgagg agccgtaatt tttcgttcgc 2340
gccgtgcggc catcaaaatg tatggatgca aatgattata catggggatg tatgggctaa 2400
atgtacgggc gacagtcaca tcatgcccct gagctgcgca cgtcaagact gtcaaggagg 2460
gtattctggg cctccatgtc gctggccggg tgacctaact tcgtataatg tatgctatac 2520
gaagttatta ggcggccgcg gcgcgccgaa ggaaaatgag aaatatcgag ggagacgatt 2580
cagaggagca ggacaaacta taaccgactg tttgttggag gatgccgtac ataacgaaca 2640
ctgctgaagc taccatgtct acagtttaga ggaatgggta caactcacag gcgagggatg 2700
gtgttcactc gtgctagcaa acgcggtggg agcaaaaagt agaatattat cttttattcg 2760
tgaaacttcg aacactgtca tctaaagatg ctatatacta atataggcat acttgataat 2820
gaaaactata aatcgtaaag acataagaga tccgcggtca caatctagct ggaataaaag 2880
attcttcagc tggagcttca gacttttgca acaaggtttc aacacctttt tgaattctag 2940
cagccaaagc tggagaaacc aacttccagg tttcgtaaac ttgcttttga atttcggctc 3000
tggtttgagc aacagattca accatgtttt ggaccatggt atcttggaat tcttgacctc 3060
taaaatctgg caaaacttcg tagaaatctc tggcgaactt gtaatcatgt tcagtagttt 3120
gcaaccagga cttggattgg atttctttca accattcatt atactttgga tctggttctc 3180
taggttcggc aaatctaact ggttgttgtt gagtcaagaa acctggatga gcaccataat 3240
tagcatcaaa agtaccttga ccatctctag caattgggtt aaaggtatga gcggatctat 3300
taactgggat ttgttgatgg ttagcaccca atctatatct ttgagcgtct gggtaagcga 3360
aaattctagc ttgtaaaatt ggatcttcag atggagcaac acctggaacc atgttagatg 3420
gagagtaagc caattgttca atttcggcga agtagttagc tgggtttctt ttcaaagtca 3480
acttaccgaa aacctttggt gggatcaaag taacatcttt tgggtaagta cccaagttcc 3540
agtgcttagt aacatccaaa atgttgtaac ccaagtttgg agcatctttt ggatcaacaa 3600
cttgaacaga agcagtccaa gttgggtatt cacctctttc aatagcttca tacaagtctc 3660
tagtagcgtg atctggatcc aaatcatttg tacctttagc ttgttgacca tcggtgaaat 3720
ttggacctct atcagatgac aagaagatgt ggacgtactt aaaagaacca tctggcttaa 3780
cccatttgta agcatgagcc atataaccag acatggatct ccaattgaac atagtaccga 3840
aatcagaaaa ttgcaacaag accatgtgca aggattcatg attgttggta acccaatccc 3900
agtacatgtt tgggttcaac aaattagttt gtggatctct tctttgagcg tgcatcaaag 3960
atgggaactt gactggatca cgaatgaaga agaatgggaa attcaacatg acccaatccc 4020
aattaccttc agtagtgtag aacttacaag ccataccttt caaatctctc ataccttcgt 4080
tagaacctct ttccaaacca gtggtagaga atctagtaac acatggagtc tttttaccga 4140
caccaaccaa catatcgata tcgcaaatat cggagatatc ggcagtaact tcaaattcac 4200
cataagcacc agcaccttta gcgtgaacaa ctctttctgg aatcttttct ctgttgaaat 4260
gggccaaggt atcaaccaaa tgatgatcag acaaagccaa agttctacca tcagctggtg 4320
gaccttcagg cttatgaaat ggacaaccat tagcagtagt gtaagttggc ttagaatctg 4380
gagccatttt aagctttgtt ttatatttgt tgtaaaaagt agataattac ttccttgatg 4440
atctgtaaaa aagagaaaaa gaaagcatct aagaacttga aaaactacga attagaaaag 4500
accaaatatg tatttcttgc attgaccaat ttatgcaagt ttatatatat gtaaatgtaa 4560
gtttcacgag gttctactaa actaaaccac ccccttggtt agaagaaaag agtgtgtgag 4620
aacaggctgt tgttgtcaca cgattcggac aattctgttt gaaagagaga gagtaacagt 4680
acgatcgaac gaactttgct ctggagatca cagtgggcat catagcatgt ggtactaaac 4740
cctttcccgc cattccagaa ccttcgattg cttgttacaa aacctgtgag ccgtcgctag 4800
gaccttgttg tgtgacgaaa ttggaagctg caatcaatag gaagacagga agtcgagcgt 4860
gtctgggttt tttcagtttt gttctttttg caaacaaatc acgagcgacg gtaatttctt 4920
tctcgataag aggccacgtg ctttatgagg gtaacatcaa ttcaagaagg agggaaacac 4980
ttcctttttc tggccctgat aatagtatga gggtgaagcc aaaataaagg attcgcgccc 5040
aaatcggcat ctttaaatgc aggtatgcga tagttcctca ctctttcctt actcacgagt 5100
aattcttgca aatgcctatt atgcagatgt tataatatct gtgcgtacgc gcccagttcg 5160
agtttatcat tatcaatact gccatttcaa agaatacgta aataattaat agtagtgatt 5220
ttcctaactt tatttagtca aaaaattagc cttttaattc tgctgtaacc cgtacatgcc 5280
caaaataggg ggcgggttac acagaatata taacatcgta ggtgtctggg tgaacagttt 5340
attcctggca tccactaaat ataatggagc ccgcttttta agctggcatc cagaaaaaaa 5400
aagaatccca gcaccaaaat attgttttct tcaccaacca tcagttcata ggtccattct 5460
cttagcgcaa ctacagagaa caggggcaca aacaggcaaa aaacggacac aacctcaatg 5520
gagtgatgca acctgcctgg agtaaatgat gacacaaggc aattgaccca cgcatgtatc 5580
tatctcattt tcttacacct tctattacct tctgctctct ctgatttgga aaaagctgaa 5640
aaaaaaggtt gaaaccagtt ccctgaaatt attcccctac ttgactaata agtatataaa 5700
gacggtaggt attgattgta attctgtaaa tctatttctt aaacttctta aattctactt 5760
ttatagttag tctttttttt agttttaaaa caccaagaac ttagtttcga ataaacacac 5820
ataaacaaac aaaaagctta aaatgggtca atccagaggt attttgggtg gtgttagaca 5880
attgatcttg gttattttgg ttggtgccta cttgtctaga ttgtctgctg ttgatgatga 5940
tagacatgat tgcagatgta gaccaggtga accatgttgg ccaactgttt ctcattggtc 6000
tactttgaac gaatctattg gtggtgcttt ggctcatgtt aagccaattg ctcatgtttg 6060
tcatcaatcc ggtcaagatt cttctgcttg cgaacaagtt ttacaagaat ccttggattc 6120
taagtggcgt gcttctcata ctggtgcttt acaagattgg gtttgggaag gtggtgctga 6180
atctaatcaa acttgttact acttgagatc aggtccagct ggttcttgtc atcaaggtag 6240
aattccatta tattccgctg ctgttaagtc cgcttctgat gttcaaaagg ttgttgattt 6300
caccagacaa cacaacttga gattggttat tagaaacacc ggtcatgatg gttctggtag 6360
atcatctggt ccagattctg ttgaaattca tacccatcac ttgaactccg ttcaatacca 6420
tccaaatttt agaccagctg gttcctctga aagacaatct gctccaggtc aaccagctgt 6480
tactgttggt gctggtattt tgttgggtga cttgtatgct agaggtgctt ctgaaggttg 6540
gatagttgtt ggtggtgaat gtccaacagt tggtgctgct ggtggttttt tacaaggtgg 6600
tggtgtttct tcttggttgt cttatgctca tggtttggct gttgataacg tcttggaata 6660
cgaagttgtt actgctaagg gtgaaatcgt tattgctaac gctcatcaaa acccagattt 6720
gttttgggct ttgagaggcg gtggtggtgg tacttttggt gttgttactc aagctacatt 6780
gcaagttcat ccagatttgc cagtttctgt tgctgatgct gttgttactg gttctagagc 6840
tgatgctact ttttggtcac atggtgttgc tgctttgttg agagctttac aattcttgaa 6900
caaccatggt actgccggtc aattcatttt gagaaacact gatgatgata ccgtccaagc 6960
tagtttgact atgtacttct ctaacttgac tgttccagct gttgctgacg aaagaatgga 7020
tcctttgaga agagctttgg aacataatgg tcatccatac caattgacct ctagattctt 7080
gccacaaatc tcttctactt tcagacatac cgctgataga tacccagaag attacggtat 7140
tttgatgggt tccgttttgg tttccttgga cttgtttaat tctgctactg gtccagctgc 7200
tttggcacaa cattttgcta gattgccaat gactccagat gatttgttgt tcacttctaa 7260
cttgggtggt agagtctctt cattgaatag agatccagct tctactgcta tgcatccagg 7320
ttggagagat gctgctcaat tattgaattt cgttagaggt gttggtgctc catctttggc 7380
tgctaaagct actgctttac atgaattgca aaccgttcaa atggccagat tatacgaaat 7440
tgaaccagcc ttccaaatct cctacagaaa tttgggtgat ccatccgaaa gaagatccag 7500
agaagtttat tggggttcta actacgctag attggttgaa gttaagagaa gatgggatcc 7560
agaaggtttg ttcttctcca aattgggtat tgatggtgat gcttgggatg ctgaaggtat 7620
gtgtagacaa acaagacaag gtgcttggaa tgttgctgtt aagtgggttc aatctttctt 7680
gggttctttg tctgcttccg ttgtttgacc gcggcacgtc cgacggcggc ccgacgggtc 7740
cgaggcctcg gagatccgtc ccccttttcc tttgtcgata tcatgtaatt agttatgtca 7800
cgcttacatt cacgccctcc ccccacatcc gctctaaccg aaaaggaagg agttagacaa 7860
cctgaagtct aggtccctat ttattttttt atagttatgt tagtattaag aacgttattt 7920
atatttcaaa tttttctttt ttttctgtac agacgcgcaa tggaaggtcg ggatgagcat 7980
atacaagcac taagaagaac aatacagaac tctacacggt attattgtgc tacaagctcg 8040
agtaaaaccg agtgttttga cgatactaac gttgttaaga aagtaacttg ttatcaaact 8100
cattaccaac ttgtgattaa ttggtgaata atatgataat tgtcgaaatt ccattgttgg 8160
taaagcctat aatattatgt atacagatta tactagaaat tctctcgaga atataagaat 8220
ccccaaaatt gaatcggtat ttctacatac taatattacc attacttctc ctttcgtttt 8280
atatgtttca ttcctattac attatcgatc tttgcatttc agcttccatt atatttgatg 8340
tctgttttat gtccccacgt tacaccgcat gtgacagtat actagtaaca tgagtgctac 8400
cgaatagatg acattttaga ctttcattcc aacaacttgg ttgacagaat gttacgtacc 8460
ctatatctaa tctatatgag gcctgaatct aactgaaagg tggaatttca gtaatttatc 8520
aagctttaat aagtttgggt agtttaactg tgcaaaaagg tatttacctt acatactgaa 8580
tcttgtctgt ttggtagcgg ctgctttatc ctgcagggca tgcaagcttg gcgtaatcat 8640
ggtcatagct gtttcctgtg tgaaattgtt atccgctcac aattccacac aacatacgag 8700
ccggaagcat aaagtgtaaa gcctggggtg cctaatgagt gagctaactc acattaattg 8760
cgttgcgctc actgcccgct ttccagtcgg gaaacctgtc gtgccagctg cattaatgaa 8820
tcggccaacg cgcggggaga ggcggtttgc gtattgggcg ctcttccgct tcctcgctca 8880
ctgactcgct gcgctcggtc gttcggctgc ggcgagcggt atcagctcac tcaaaggcgg 8940
taatacggtt atccacagaa tcaggggata acgcaggaaa gaacatgtga gcaaaaggcc 9000
agcaaaaggc caggaaccgt aaaaaggccg cgttgctggc gtttttccat aggctccgcc 9060
cccctgacga gcatcacaaa aatcgacgct caagtcagag gtggcgaaac ccgacaggac 9120
tataaagata ccaggcgttt ccccctggaa gctccctcgt gcgctctcct gttccgaccc 9180
tgccgcttac cggatacctg tccgcctttc tcccttcggg aagcgtggcg ctttctcata 9240
gctcacgctg taggtatctc agttcggtgt aggtcgttcg ctccaagctg ggctgtgtgc 9300
acgaaccccc cgttcagccc gaccgctgcg ccttatccgg taactatcgt cttgagtcca 9360
acccggtaag acacgactta tcgccactgg cagcagccac tggtaacagg attagcagag 9420
cgaggtatgt aggcggtgct acagagttct tgaagtggtg gcctaactac ggctacacta 9480
gaaggacagt atttggtatc tgcgctctgc tgaagccagt taccttcgga aaaagagttg 9540
gtagctcttg atccggcaaa caaaccaccg ctggtagcgg tggttttttt gtttgcaagc 9600
agcagattac gcgcagaaaa aaaggatctc aagaagatcc tttgatcttt tctacggggt 9660
ctgacgctca gtggaacgaa aactcacgtt aagggatttt ggtcatgaga ttatcaaaaa 9720
ggatcttcac ctagatcctt ttaaattaaa aatgaagttt taaatcaatc taaagtatat 9780
atgagtaaac ttggtctgac agttaccaat gcttaatcag tgaggcacct atctcagcga 9840
tctgtctatt tcgttcatcc atagttgcct gactccccgt cgtgtagata actacgatac 9900
gggagggctt accatctggc cccagtgctg caatgatacc gcgagaccca cgctcaccgg 9960
ctccagattt atcagcaata aaccagccag ccggaagggc cgagcgcaga agtggtcctg 10020
caactttatc cgcctccatc cagtctatta attgttgccg ggaagctaga gtaagtagtt 10080
cgccagttaa tagtttgcgc aacgttgttg ccattgctac aggcatcgtg gtgtcacgct 10140
cgtcgtttgg tatggcttca ttcagctccg gttcccaacg atcaaggcga gttacatgat 10200
cccccatgtt gtgcaaaaaa gcggttagct ccttcggtcc tccgatcgtt gtcagaagta 10260
agttggccgc agtgttatca ctcatggtta tggcagcact gcataattct cttactgtca 10320
tgccatccgt aagatgcttt tctgtgactg gtgagtactc aaccaagtca ttctgagaat 10380
agtgtatgcg gcgaccgagt tgctcttgcc cggcgtcaat acgggataat accgcgccac 10440
atagcagaac tttaaaagtg ctcatcattg gaaaacgttc ttcggggcga aaactctcaa 10500
ggatcttacc gctgttgaga tccagttcga tgtaacccac tcgtgcaccc aactgatctt 10560
cagcatcttt tactttcacc agcgtttctg ggtgagcaaa aacaggaagg caaaatgccg 10620
caaaaaaggg aataagggcg acacggaaat gttgaatact catactcttc ctttttcaat 10680
attattgaag catttatcag ggttattgtc tcatgagcgg atacatattt gaatgtattt 10740
agaaaaataa acaaataggg gttccgcgca catttccccg aaaagtgcca cctgacgtct 10800
aagaaaccat tattatcatg acattaacct ataaaaatag gcgtatcacg aggccctttc 10860
gtctcgcgcg tttcggtgat gacggtgaaa acctctgaca catgcagctc ccggagacgg 10920
tcacagcttg tctgtaagcg gatgccggga gcagacaagc ccgtcagggc gcgtcagcgg 10980
gtgttggcgg gtgtcggggc tggcttaact atgcggcatc agagcagatt gtactgagag 11040
tgcaccatat gcggtgtgaa ataccgcaca gatgcgtaag gagaaaatac cgcatcaggc 11100
gccattcgcc attcaggctg cgcaactgtt gggaagggcg at 11142
<210> 116
<211> 7110
<212> DNA
<213> Artificial Sequence
<220>
<223> pEVE2342 (YORW?22-BleR-P/A+Aj_EasD)
<400> 116
cggtgcgggc ctcttcgcta ttacgccagc tggcgaaagg gggatgtgct gcaaggcgat 60
taagttgggt aacgccaggg ttttcccagt cacgacgttg taaaacgacg gccagtgaat 120
tcgagctccc tgcaggcacc ggagcttgga tatgataaac gaaatattct tgaatcgtga 180
gatcgcctgt tttcaaaacc gttggaggca gaaacaattt tgtcacaaga tgggcattct 240
accccatcct tgctgtatta ttgtagtctc gctttctttt atgctggaca aatgagacta 300
ctgcacattt ttatacgttc ttggtttttt ttaaaggtgt ggtttcggca ttatcctgcc 360
gcacgtttct tggataattc atcctgattc tctattttaa acgcttcagc ctatcaggat 420
ttggttttga tacatactgc aagagtgtat ctcgggaaca gtcatttatt ccgcaacaaa 480
cttaattgcg gaacgcgtta ggcgatttct agcatatatc aaataccgtt cgcgatttct 540
tctgggttcg tctcttttct tttaaatact tattaacgta ctcaaacaac tacacttcgt 600
tgtatctcag aatgagatcc ctcagtatga caatacatca ttctaaacgt tcgtaaaaca 660
catatgaaac aactttataa caaagcgaac aaaatgggca acatgagatg aaactccgcg 720
tcccttagct gaactaccca aacgtacgaa tgcctgaaca attagtttag atccgagatt 780
ccgcgcttcc atcatttagt ataatccata ttttatataa tatataggat aagtaacagc 840
ccgcgaagcg gccgccagct gaagcttcgt acgctgcagg tcgacaaccc ttaatataac 900
ttcgtataat gtatgctata cgaagttatt aggtctagag atctgtttag cttgcctcgt 960
ccccgccggg tcacccggcc agcgacatgg aggcccagaa taccctcctt gacagtcttg 1020
acgtgcgcag ctcaggggca tgatgtgact gtcgcccgta catttagccc atacatcccc 1080
atgtataatc atttgcatcc atacattttg atggccgcac ggcgcgaagc aaaaattacg 1140
gctcctcgct gcagacctgc gagcagggaa acgctcccct cacagacgcg ttgaattgtc 1200
cccacgccgc gcccctgtag agaaatataa aaggttagga tttgccactg aggttcttct 1260
ttcatatact tccttttaaa atcttgctag gatacagttc tcacatcaca tccgaacata 1320
aacaaccatg ggtatgaccg accaagcgac gcccaacctg ccatcacgag atttcgatcc 1380
caccgccgcc ttctatgaaa ggttgggctt cggaatcgtt ttccgggacg ccggctggat 1440
gatcctccag cgcggggatc tcatgctgga gttcttcgcc caccccgggc tcgatcccct 1500
cgcgagttgg ttcagctgct gcctgaggct ggacgacctc gcggagttct accggcagtg 1560
caaatccgtc ggcatccagg aaaccagcag cggctatccg cgcatccatg cccccgaact 1620
gcaggagtgg ggaggcacga tggccgcttt ggtcgacccg gacgggacgc tcctgcgcct 1680
gatacagaac gaattgcttg caggcatctc atgatcagta ctgacaataa aaagattctt 1740
gttttcaaga acttgtcatt tgtatagttt ttttatattg tagttgttct attttaatca 1800
aatgttagcg tgatttatat tttttttcgc ctcgacatca tctgcccaga tgcgaagtta 1860
agtgcgcaga aagtaatatc atgcgtcaat cgtatgtgaa tgctggtcgc tatactgctg 1920
tcgattcgat actaacgccg ccatccagtg tcgaaaacga gctctcgaga acccttaata 1980
taacttcgta taatgtatgc tatacgaagt tattaggtga tatcagatcc actagtggcc 2040
tatgcggccg cggcgcgtga aggaaaatga gaaatatcga gggagacgat tcagaggagc 2100
aggacaaact ataaccgact gtttgttgga ggatgccgta cataacgaac actgctgaag 2160
ctaccatgtc tacagtttag aggaatgggt acaactcaca ggcgagggat ggtgttcact 2220
cgtgctagca aacgcggtgg gagcaaaaag tagaatatta tcttttattc gtgaaacttc 2280
gaacactgtc atctaaagat gctatatact aatataggca tacttgataa tgaaaactat 2340
aaatcgtaaa gacataagag atccgcggtt atggagttgc agcaccaacg ttaatattgg 2400
caccgtaaac tggtctagaa tcttcagaca acaaccagac gatagttcta gcaacatctt 2460
caggttgaat gattgggaaa ccttctttga tgtaggcttc ttttacttcg tcgatagatt 2520
tagcttgtgg catgaattgt ggcaaagctg gagtatcggt aatacctgga gaaacagaat 2580
tgattctaat acccaatggc caagcatcca tagcagcaca agaagtaaag taagcacaag 2640
cacctttaga agtaccgtaa gcataagcat ctggcaaatg gataaaggca gcaatggaac 2700
caatgttaac gatagctcta tcttttggac ctggcaaagc cttcatagct ctaataccag 2760
ctctagtaca gtagaaaaca ccgtccaagt taaccttgaa aactctttgc cattcttcat 2820
cagtttcttc caacaaagct ggagcttgtc tagcaccagc tggttgagca ataccagcaa 2880
cattagccaa accatgcaag tcaccaaaag tttgaacgat agtttgcaac caagcatcaa 2940
cttcagcgga tgaagaaaca tccaatctag tgcaatgaac ctttgtagct ggattcaagg 3000
cttgaatatc agaggtaacg gattcgaaac cctttggaga aatatcacca atgcatataa 3060
cagcagcacc tctagcagcc aataatctac aagtagcagc accaatacca gaagcaccac 3120
cagtaacagc gaaaattcta gagttgacaa cggtcatttt aagctttgtt ttatatttgt 3180
tgtaaaaagt agataattac ttccttgatg atctgtaaaa aagagaaaaa gaaagcatct 3240
aagaacttga aaaactacga attagaaaag accaaatatg tatttcttgc attgaccaat 3300
ttatgcaagt ttatatatat gtaaatgtaa gtttcacgag gttctactaa actaaaccac 3360
ccccttggtt agaagaaaag agtgtgtgag aacaggctgt tgttgtcaca cgattcggac 3420
aattctgttt gaaagagaga gagtaacagt acgatcgaac gaactttgct ctggagatca 3480
cagtgggcat catagcatgt ggtactaaac cctttcccgc cattccagaa ccttcgattg 3540
cttgttacaa aacctgtgag ccgtcgctag gaccttgttg tgtgacgaaa ttggaagctg 3600
caatcaatag gaagacagga agtcgagcgt gtctgggttt tttcagtttt gttctttttg 3660
caaacaaatc acgagcgacg gtaatttctt tctcgataag aggccacgtg ctttatgagg 3720
gtaacatcaa ttcaagaagg agggaaacac ttcctttttc tggccctgat aatagtatga 3780
gggtgaagcc aaaataaagg attcgcgccc aaatcggcat ctttaaatgc aggtatgcga 3840
tagttcctca ctctttcctt actcacgagt aattcttgca aatgcctatt atgcagatgt 3900
tataatatct gtgcgtggcg cgccggacca actatcatcc gctaattact gacattacca 3960
aatgagatct gtgaatgggc aagataaaaa acaaaaattg aaatgtttga cgttatgtaa 4020
aactattaat tccttcgctt tcggcggtca cagaatttgc gtgtagctga ctcttgttca 4080
atcaatatca tttgttactt tatttgaaag tctgtattac tgcgcctatt gtcatccgta 4140
ccaaagaacg tcaaaaagaa acaagataat ttttgtgctt acaccattta tagatcactg 4200
agcccagaat atcgctggag ctcagtgtaa gtggcatgaa cacaactctg actgatcgca 4260
catattgccg ttatcataaa tactagttgt acttgtcaat gcgacgaatg gcatcatgcc 4320
tattattacg ttcctctttt tccgtttcat gtttccagaa tgctattgaa tctaacactt 4380
caattataaa aaagaataaa tccgcaataa ttttaggcta attgttgtac tgtcaagcga 4440
acctaatggt taaaattcag aggaaccttc gacgtagtct gatcgctact tctatatctt 4500
atgttcccag tcaatcaaaa gttgatacta taatagctgc catttatacc tgttagttat 4560
ggcgatcgtt tatcacgcct gcagggcatg caagcttggc gtaatcatgg tcatagctgt 4620
ttcctgtgtg aaattgttat ccgctcacaa ttccacacaa catacgagcc ggaagcataa 4680
agtgtaaagc ctggggtgcc taatgagtga gctaactcac attaattgcg ttgcgctcac 4740
tgcccgcttt ccagtcggga aacctgtcgt gccagctgca ttaatgaatc ggccaacgcg 4800
cggggagagg cggtttgcgt attgggcgct cttccgcttc ctcgctcact gactcgctgc 4860
gctcggtcgt tcggctgcgg cgagcggtat cagctcactc aaaggcggta atacggttat 4920
ccacagaatc aggggataac gcaggaaaga acatgtgagc aaaaggccag caaaaggcca 4980
ggaaccgtaa aaaggccgcg ttgctggcgt ttttccatag gctccgcccc cctgacgagc 5040
atcacaaaaa tcgacgctca agtcagaggt ggcgaaaccc gacaggacta taaagatacc 5100
aggcgtttcc ccctggaagc tccctcgtgc gctctcctgt tccgaccctg ccgcttaccg 5160
gatacctgtc cgcctttctc ccttcgggaa gcgtggcgct ttctcatagc tcacgctgta 5220
ggtatctcag ttcggtgtag gtcgttcgct ccaagctggg ctgtgtgcac gaaccccccg 5280
ttcagcccga ccgctgcgcc ttatccggta actatcgtct tgagtccaac ccggtaagac 5340
acgacttatc gccactggca gcagccactg gtaacaggat tagcagagcg aggtatgtag 5400
gcggtgctac agagttcttg aagtggtggc ctaactacgg ctacactaga aggacagtat 5460
ttggtatctg cgctctgctg aagccagtta ccttcggaaa aagagttggt agctcttgat 5520
ccggcaaaca aaccaccgct ggtagcggtg gtttttttgt ttgcaagcag cagattacgc 5580
gcagaaaaaa aggatctcaa gaagatcctt tgatcttttc tacggggtct gacgctcagt 5640
ggaacgaaaa ctcacgttaa gggattttgg tcatgagatt atcaaaaagg atcttcacct 5700
agatcctttt aaattaaaaa tgaagtttta aatcaatcta aagtatatat gagtaaactt 5760
ggtctgacag ttaccaatgc ttaatcagtg aggcacctat ctcagcgatc tgtctatttc 5820
gttcatccat agttgcctga ctccccgtcg tgtagataac tacgatacgg gagggcttac 5880
catctggccc cagtgctgca atgataccgc gagacccacg ctcaccggct ccagatttat 5940
cagcaataaa ccagccagcc ggaagggccg agcgcagaag tggtcctgca actttatccg 6000
cctccatcca gtctattaat tgttgccggg aagctagagt aagtagttcg ccagttaata 6060
gtttgcgcaa cgttgttgcc attgctacag gcatcgtggt gtcacgctcg tcgtttggta 6120
tggcttcatt cagctccggt tcccaacgat caaggcgagt tacatgatcc cccatgttgt 6180
gcaaaaaagc ggttagctcc ttcggtcctc cgatcgttgt cagaagtaag ttggccgcag 6240
tgttatcact catggttatg gcagcactgc ataattctct tactgtcatg ccatccgtaa 6300
gatgcttttc tgtgactggt gagtactcaa ccaagtcatt ctgagaatag tgtatgcggc 6360
gaccgagttg ctcttgcccg gcgtcaatac gggataatac cgcgccacat agcagaactt 6420
taaaagtgct catcattgga aaacgttctt cggggcgaaa actctcaagg atcttaccgc 6480
tgttgagatc cagttcgatg taacccactc gtgcacccaa ctgatcttca gcatctttta 6540
ctttcaccag cgtttctggg tgagcaaaaa caggaaggca aaatgccgca aaaaagggaa 6600
taagggcgac acggaaatgt tgaatactca tactcttcct ttttcaatat tattgaagca 6660
tttatcaggg ttattgtctc atgagcggat acatatttga atgtatttag aaaaataaac 6720
aaataggggt tccgcgcaca tttccccgaa aagtgccacc tgacgtctaa gaaaccatta 6780
ttatcatgac attaacctat aaaaataggc gtatcacgag gccctttcgt ctcgcgcgtt 6840
tcggtgatga cggtgaaaac ctctgacaca tgcagctccc ggagacggtc acagcttgtc 6900
tgtaagcgga tgccgggagc agacaagccc gtcagggcgc gtcagcgggt gttggcgggt 6960
gtcggggctg gcttaactat gcggcatcag agcagattgt actgagagtg caccatatgc 7020
ggtgtgaaat accgcacaga tgcgtaagga gaaaataccg catcaggcgc cattcgccat 7080
tcaggctgcg caactgttgg gaagggcgat 7110
<210> 117
<211> 8932
<212> DNA
<213> Artificial Sequence
<220>
<223> pEVE2343 (YORW?22-BleR-G/C+Aj_EasH-P/A+Aj_EasD)
<400> 117
cggtgcgggc ctcttcgcta ttacgccagc tggcgaaagg gggatgtgct gcaaggcgat 60
taagttgggt aacgccaggg ttttcccagt cacgacgttg taaaacgacg gccagtgaat 120
tcgagctccc tgcaggcacc ggagcttgga tatgataaac gaaatattct tgaatcgtga 180
gatcgcctgt tttcaaaacc gttggaggca gaaacaattt tgtcacaaga tgggcattct 240
accccatcct tgctgtatta ttgtagtctc gctttctttt atgctggaca aatgagacta 300
ctgcacattt ttatacgttc ttggtttttt ttaaaggtgt ggtttcggca ttatcctgcc 360
gcacgtttct tggataattc atcctgattc tctattttaa acgcttcagc ctatcaggat 420
ttggttttga tacatactgc aagagtgtat ctcgggaaca gtcatttatt ccgcaacaaa 480
cttaattgcg gaacgcgtta ggcgatttct agcatatatc aaataccgtt cgcgatttct 540
tctgggttcg tctcttttct tttaaatact tattaacgta ctcaaacaac tacacttcgt 600
tgtatctcag aatgagatcc ctcagtatga caatacatca ttctaaacgt tcgtaaaaca 660
catatgaaac aactttataa caaagcgaac aaaatgggca acatgagatg aaactccgcg 720
tcccttagct gaactaccca aacgtacgaa tgcctgaaca attagtttag atccgagatt 780
ccgcgcttcc atcatttagt ataatccata ttttatataa tatataggat aagtaacagc 840
ccgcgaagcg gccgccagct gaagcttcgt acgctgcagg tcgacaaccc ttaatataac 900
ttcgtataat gtatgctata cgaagttatt aggtctagag atctgtttag cttgcctcgt 960
ccccgccggg tcacccggcc agcgacatgg aggcccagaa taccctcctt gacagtcttg 1020
acgtgcgcag ctcaggggca tgatgtgact gtcgcccgta catttagccc atacatcccc 1080
atgtataatc atttgcatcc atacattttg atggccgcac ggcgcgaagc aaaaattacg 1140
gctcctcgct gcagacctgc gagcagggaa acgctcccct cacagacgcg ttgaattgtc 1200
cccacgccgc gcccctgtag agaaatataa aaggttagga tttgccactg aggttcttct 1260
ttcatatact tccttttaaa atcttgctag gatacagttc tcacatcaca tccgaacata 1320
aacaaccatg ggtatgaccg accaagcgac gcccaacctg ccatcacgag atttcgatcc 1380
caccgccgcc ttctatgaaa ggttgggctt cggaatcgtt ttccgggacg ccggctggat 1440
gatcctccag cgcggggatc tcatgctgga gttcttcgcc caccccgggc tcgatcccct 1500
cgcgagttgg ttcagctgct gcctgaggct ggacgacctc gcggagttct accggcagtg 1560
caaatccgtc ggcatccagg aaaccagcag cggctatccg cgcatccatg cccccgaact 1620
gcaggagtgg ggaggcacga tggccgcttt ggtcgacccg gacgggacgc tcctgcgcct 1680
gatacagaac gaattgcttg caggcatctc atgatcagta ctgacaataa aaagattctt 1740
gttttcaaga acttgtcatt tgtatagttt ttttatattg tagttgttct attttaatca 1800
aatgttagcg tgatttatat tttttttcgc ctcgacatca tctgcccaga tgcgaagtta 1860
agtgcgcaga aagtaatatc atgcgtcaat cgtatgtgaa tgctggtcgc tatactgctg 1920
tcgattcgat actaacgccg ccatccagtg tcgaaaacga gctctcgaga acccttaata 1980
taacttcgta taatgtatgc tatacgaagt tattaggtga tatcagatcc actagtggcc 2040
tatgcggccg cggcgcgtct gtacagaaaa aaaagaaaaa tttgaaatat aaataacgtt 2100
cttaatacta acataactat aaaaaaataa atagggacct agacttcagg ttgtctaact 2160
ccttcctttt cggttagagc ggatgtgggg ggagggcgtg aatgtaagcg tgacataact 2220
aattacatga tatcgacaaa ggaaaagggg gacggatctc cgaggcctcg gacccgtcgg 2280
gccgccgtcg gacgtgccgc ggttaggcct tcttagcaac ttgattagac ttcaagtcaa 2340
taccggtagc caaatccttc aaatcgtagg tccaaatatc aacaccctta gcagaaacag 2400
ttctccaacc aaccattttt tgggccaatg gagtcatagt ttcaaccaat tctcttggca 2460
aagcaacact agcttcaact ggagtgaatt gacaagattg catgatcaat gggaaacctc 2520
ttctcaaaaa atcagtagtg ttgttcttac cggcaccatg taaagtttta ccagtaaaca 2580
aaacagcatc acctggattc ataacagctg gaatagcagt atcagctttt ggtggtggag 2640
cagaaaaatc ttcccacaaa tgagaacctg gcaaaactct agtagcacca ttttcagcag 2700
tgaaatcagt caaagcagtc aagaagttca acatagattc tggagattgt ctacccaagg 2760
tagtacagat tggaatacca tcatgttctc tatgcaattc ttgaccatgg taacctggtt 2820
cagtttccaa aacagaagaa gtagtcaacc agtaatcacc agattctgga ccgaataatc 2880
tcttgcacaa accgtgcaat aattcgtcgt tcatcaattc atctctgaac aacttggaga 2940
acaaaggcaa agatgacaat tgtctcatac cagccaagta agattcttgg taggattttt 3000
gacccaattc ccattgcttc atgtgtggat caatttcacc gttgaatctt ctaacttggt 3060
cagcagacat gaaaccttcg ataacaacaa cgccatcttc ttgcaagact tggaaaatag 3120
cttcatcacc agctgatctt ggaattcttc tcaaggttgg cttagaagtt tcggtaatgg 3180
tcattttaag ctttttgttt gtttatgtgt gtttattcga aactaagttc ttggtgtttt 3240
aaaactaaaa aaaagactaa ctataaaagt agaatttaag aagtttaaga aatagattta 3300
cagaattaca atcaatacct accgtcttta tatacttatt agtcaagtag gggaataatt 3360
tcagggaact ggtttcaacc ttttttttca gctttttcca aatcagagag agcagaaggt 3420
aatagaaggt gtaagaaaat gagatagata catgcgtggg tcaattgcct tgtgtcatca 3480
tttactccag gcaggttgca tcactccatt gaggttgtgt ccgttttttg cctgtttgtg 3540
cccctgttct ctgtagttgc gctaagagaa tggacctatg aactgatggt tggtgaagaa 3600
aacaatattt tggtgctggg attctttttt tttctggatg ccagcttaaa aagcgggctc 3660
cattatattt agtggatgcc aggaataaac tgttcaccca gacacctacg atgttatata 3720
ttctgtgtaa cccgccccct attttgggca tgtacgggtt acagcagaat taaaaggcta 3780
attttttgac taaataaagt taggaaaatc actactatta attatttacg tattctttga 3840
aatggcagta ttgataatga taaactcgaa cttggcgcgt acgcacagat attataacat 3900
ctgcataata ggcatttgca agaattactc gtgagtaagg aaagagtgag gaactatcgc 3960
atacctgcat ttaaagatgc cgatttgggc gcgaatcctt tattttggct tcaccctcat 4020
actattatca gggccagaaa aaggaagtgt ttccctcctt cttgaattga tgttaccctc 4080
ataaagcacg tggcctctta tcgagaaaga aattaccgtc gctcgtgatt tgtttgcaaa 4140
aagaacaaaa ctgaaaaaac ccagacacgc tcgacttcct gtcttcctat tgattgcagc 4200
ttccaatttc gtcacacaac aaggtcctag cgacggctca caggttttgt aacaagcaat 4260
cgaaggttct ggaatggcgg gaaagggttt agtaccacat gctatgatgc ccactgtgat 4320
ctccagagca aagttcgttc gatcgtactg ttactctctc tctttcaaac agaattgtcc 4380
gaatcgtgtg acaacaacag cctgttctca cacactcttt tcttctaacc aagggggtgg 4440
tttagtttag tagaacctcg tgaaacttac atttacatat atataaactt gcataaattg 4500
gtcaatgcaa gaaatacata tttggtcttt tctaattcgt agtttttcaa gttcttagat 4560
gctttctttt tctctttttt acagatcatc aaggaagtaa ttatctactt tttacaacaa 4620
atataaaaca aagcttaaaa tgaccgttgt caactctaga attttcgctg ttactggtgg 4680
tgcttctggt attggtgctg ctacttgtag attattggct gctagaggtg ctgctgttat 4740
atgcattggt gatatttctc caaagggttt cgaatccgtt acctctgata ttcaagcctt 4800
gaatccagct acaaaggttc attgcactag attggatgtt tcttcatccg ctgaagttga 4860
tgcttggttg caaactatcg ttcaaacttt tggtgacttg catggtttgg ctaatgttgc 4920
tggtattgct caaccagctg gtgctagaca agctccagct ttgttggaag aaactgatga 4980
agaatggcaa agagttttca aggttaactt ggacggtgtt ttctactgta ctagagctgg 5040
tattagagct atgaaggctt tgccaggtcc aaaagataga gctatcgtta acattggttc 5100
cattgctgcc tttatccatt tgccagatgc ttatgcttac ggtacttcta aaggtgcttg 5160
tgcttacttt acttcttgtg ctgctatgga tgcttggcca ttgggtatta gaatcaattc 5220
tgtttctcca ggtattaccg atactccagc tttgccacaa ttcatgccac aagctaaatc 5280
tatcgacgaa gtaaaagaag cctacatcaa agaaggtttc ccaatcattc aacctgaaga 5340
tgttgctaga actatcgtct ggttgttgtc tgaagattct agaccagttt acggtgccaa 5400
tattaacgtt ggtgctgcaa ctccataacc gcggatctct tatgtcttta cgatttatag 5460
ttttcattat caagtatgcc tatattagta tatagcatct ttagatgaca gtgttcgaag 5520
tttcacgaat aaaagataat attctacttt ttgctcccac cgcgtttgct agcacgagtg 5580
aacaccatcc ctcgcctgtg agttgtaccc attcctctaa actgtagaca tggtagcttc 5640
agcagtgttc gttatgtacg gcatcctcca acaaacagtc ggttatagtt tgtcctgctc 5700
ctctgaatcg tctccctcga tatttctcat tttccttcgg cgcgccggac caactatcat 5760
ccgctaatta ctgacattac caaatgagat ctgtgaatgg gcaagataaa aaacaaaaat 5820
tgaaatgttt gacgttatgt aaaactatta attccttcgc tttcggcggt cacagaattt 5880
gcgtgtagct gactcttgtt caatcaatat catttgttac tttatttgaa agtctgtatt 5940
actgcgccta ttgtcatccg taccaaagaa cgtcaaaaag aaacaagata atttttgtgc 6000
ttacaccatt tatagatcac tgagcccaga atatcgctgg agctcagtgt aagtggcatg 6060
aacacaactc tgactgatcg cacatattgc cgttatcata aatactagtt gtacttgtca 6120
atgcgacgaa tggcatcatg cctattatta cgttcctctt tttccgtttc atgtttccag 6180
aatgctattg aatctaacac ttcaattata aaaaagaata aatccgcaat aattttaggc 6240
taattgttgt actgtcaagc gaacctaatg gttaaaattc agaggaacct tcgacgtagt 6300
ctgatcgcta cttctatatc ttatgttccc agtcaatcaa aagttgatac tataatagct 6360
gccatttata cctgttagtt atggcgatcg tttatcacgc ctgcagggca tgcaagcttg 6420
gcgtaatcat ggtcatagct gtttcctgtg tgaaattgtt atccgctcac aattccacac 6480
aacatacgag ccggaagcat aaagtgtaaa gcctggggtg cctaatgagt gagctaactc 6540
acattaattg cgttgcgctc actgcccgct ttccagtcgg gaaacctgtc gtgccagctg 6600
cattaatgaa tcggccaacg cgcggggaga ggcggtttgc gtattgggcg ctcttccgct 6660
tcctcgctca ctgactcgct gcgctcggtc gttcggctgc ggcgagcggt atcagctcac 6720
tcaaaggcgg taatacggtt atccacagaa tcaggggata acgcaggaaa gaacatgtga 6780
gcaaaaggcc agcaaaaggc caggaaccgt aaaaaggccg cgttgctggc gtttttccat 6840
aggctccgcc cccctgacga gcatcacaaa aatcgacgct caagtcagag gtggcgaaac 6900
ccgacaggac tataaagata ccaggcgttt ccccctggaa gctccctcgt gcgctctcct 6960
gttccgaccc tgccgcttac cggatacctg tccgcctttc tcccttcggg aagcgtggcg 7020
ctttctcata gctcacgctg taggtatctc agttcggtgt aggtcgttcg ctccaagctg 7080
ggctgtgtgc acgaaccccc cgttcagccc gaccgctgcg ccttatccgg taactatcgt 7140
cttgagtcca acccggtaag acacgactta tcgccactgg cagcagccac tggtaacagg 7200
attagcagag cgaggtatgt aggcggtgct acagagttct tgaagtggtg gcctaactac 7260
ggctacacta gaaggacagt atttggtatc tgcgctctgc tgaagccagt taccttcgga 7320
aaaagagttg gtagctcttg atccggcaaa caaaccaccg ctggtagcgg tggttttttt 7380
gtttgcaagc agcagattac gcgcagaaaa aaaggatctc aagaagatcc tttgatcttt 7440
tctacggggt ctgacgctca gtggaacgaa aactcacgtt aagggatttt ggtcatgaga 7500
ttatcaaaaa ggatcttcac ctagatcctt ttaaattaaa aatgaagttt taaatcaatc 7560
taaagtatat atgagtaaac ttggtctgac agttaccaat gcttaatcag tgaggcacct 7620
atctcagcga tctgtctatt tcgttcatcc atagttgcct gactccccgt cgtgtagata 7680
actacgatac gggagggctt accatctggc cccagtgctg caatgatacc gcgagaccca 7740
cgctcaccgg ctccagattt atcagcaata aaccagccag ccggaagggc cgagcgcaga 7800
agtggtcctg caactttatc cgcctccatc cagtctatta attgttgccg ggaagctaga 7860
gtaagtagtt cgccagttaa tagtttgcgc aacgttgttg ccattgctac aggcatcgtg 7920
gtgtcacgct cgtcgtttgg tatggcttca ttcagctccg gttcccaacg atcaaggcga 7980
gttacatgat cccccatgtt gtgcaaaaaa gcggttagct ccttcggtcc tccgatcgtt 8040
gtcagaagta agttggccgc agtgttatca ctcatggtta tggcagcact gcataattct 8100
cttactgtca tgccatccgt aagatgcttt tctgtgactg gtgagtactc aaccaagtca 8160
ttctgagaat agtgtatgcg gcgaccgagt tgctcttgcc cggcgtcaat acgggataat 8220
accgcgccac atagcagaac tttaaaagtg ctcatcattg gaaaacgttc ttcggggcga 8280
aaactctcaa ggatcttacc gctgttgaga tccagttcga tgtaacccac tcgtgcaccc 8340
aactgatctt cagcatcttt tactttcacc agcgtttctg ggtgagcaaa aacaggaagg 8400
caaaatgccg caaaaaaggg aataagggcg acacggaaat gttgaatact catactcttc 8460
ctttttcaat attattgaag catttatcag ggttattgtc tcatgagcgg atacatattt 8520
gaatgtattt agaaaaataa acaaataggg gttccgcgca catttccccg aaaagtgcca 8580
cctgacgtct aagaaaccat tattatcatg acattaacct ataaaaatag gcgtatcacg 8640
aggccctttc gtctcgcgcg tttcggtgat gacggtgaaa acctctgaca catgcagctc 8700
ccggagacgg tcacagcttg tctgtaagcg gatgccggga gcagacaagc ccgtcagggc 8760
gcgtcagcgg gtgttggcgg gtgtcggggc tggcttaact atgcggcatc agagcagatt 8820
gtactgagag tgcaccatat gcggtgtgaa ataccgcaca gatgcgtaag gagaaaatac 8880
cgcatcaggc gccattcgcc attcaggctg cgcaactgtt gggaagggcg at 8932
<210> 118
<211> 9280
<212> DNA
<213> Artificial Sequence
<220>
<223> pEVE2344 (YPRC?3-NatR-G/C+Aj_EasA-P/A+Aj_EasG)
<400> 118
cggtgcgggc ctcttcgcta ttacgccagc tggcgaaagg gggatgtgct gcaaggcgat 60
taagttgggt aacgccaggg ttttcccagt cacgacgttg taaaacgacg gccagtgaat 120
tcgagctccc tgcaggaaag gaggtgcacg cattatggag accactacga tacgatagct 180
gcgttgttgt tgaaggggtt tcttaaggtt gttttcgttg aaggtaaata ttggtcgttt 240
ttgtgcagca tattgtcctc tagatgcaaa ctctgcaggt ccatttgcag taaagtgagt 300
tgcctctcga agaatcatta atttcgtata accgtcacta ttaaagtcag aaaataaatt 360
ctgtcgtaga caatgttacc ataatgttct tgtccatttt gcatacactt taaatattca 420
tttgatttct cagggttcat gatcataata aattgcgcat tcgcaaggcg gtagtattat 480
aatggggtcc atcattctgt agcaagaagt tacagtacgc tgttcaagcg ttaaacaaga 540
taagtaatct cgaatgaaac attcatattt cgcatgagcc aacatacagt tgctgagtaa 600
tcttcattgc gcttatttat cggcattgag attgtaaagg aagtaaaacg catttttgca 660
gatctgttct cttatgtatt tttaatcgtc cttgtatgga agtatcaaag gggacgttct 720
tcacctcctt ggaagcggcc gcctaataac ttcgtatagc atacattata cgaagttagg 780
tcacccggcc agcgacatgg aggcccagaa taccctcctt gacagtcttg acgtgcgcag 840
ctcaggggca tgatgtgact gtcgcccgta catttagccc atacatcccc atgtataatc 900
atttgcatcc atacattttg atggccgcac ggcgcgaacg aaaaattacg gctcctcgct 960
gcagacctgc gagcagggaa acgctcccct cacagacgcg ttgaattgtc cccacgccgc 1020
gcccctgtag agaaatataa aaggttagga tttgccactg aggttcttct ttcatatact 1080
tccttttaaa atcttgctag gatacagttc tcacatcaca tccgaacata aacaaccatg 1140
ggtaccactc ttgacgacac ggcttaccgg taccgcacca gtgtcccggg ggacgccgag 1200
gccatcgagg cactggatgg gtccttcacc accgacaccg tcttccgcgt caccgccacc 1260
ggggacggct tcaccctgcg ggaggtgccg gtggacccgc ccctgaccaa ggtgttcccc 1320
gacgacgaat cggacgacga atcggacgcc ggggaggacg gcgacccgga ctcccggacg 1380
ttcgtcgcgt acggggacga cggcgacctg gcgggcttcg tggtcgtctc gtactccggc 1440
tggaaccgcc ggctgaccgt cgaggacatc gaggtcgccc cggagcaccg ggggcacggg 1500
gtcgggcgcg cgttgatggg gctcgcgacg gagttcgccc gcgagcgggg cgccgggcac 1560
ctctggctgg aggtcaccaa cgtcaacgca ccggcgatcc acgcgtaccg gcggatgggg 1620
ttcaccctct gcggcctgga caccgccctg tacgacggca ccgcctcgga cggcgagcag 1680
gcgctctaca tgagcatgcc ctgcccctaa tcagtactga caataaaaag attcttgttt 1740
tcaagaactt gtcatttgta tagttttttt atattgtagt tgttctattt taatcaaatg 1800
ttagcgtgat ttatattttt tttgcctcga catcatctgc ccagatgcga agttaagtgc 1860
gcagaaagta atatcatgcg tcaatcgtat gtgaatgctg gtcgctatac tgctgtcgat 1920
tcgatactaa cgccgccatc cagtgtcgaa ataacttcgt atagcataca ttatacgaag 1980
ttagcggccg cggcgcgtct gtacagaaaa aaaagaaaaa tttgaaatat aaataacgtt 2040
cttaatacta acataactat aaaaaaataa atagggacct agacttcagg ttgtctaact 2100
ccttcctttt cggttagagc ggatgtgggg ggagggcgtg aatgtaagcg tgacataact 2160
aattacatga tatcgacaaa ggaaaagggg gacggatctc cgaggcctcg gacccgtcgg 2220
gccgccgtcg gacgtgccgc ggttaacctt gacaaataga accggtagca gcagcttctt 2280
tcttcttcaa tctttgaccg aattcagcaa catattcagc ggagtatgga tagtccaagt 2340
aaccttcttt caataaggag gtgtagaaag aagctctatc gtacttttgc aatggaatac 2400
ctctttgcat tctgaatggc aaatctggag tagaaataaa gtatctaccg aaggcaacag 2460
caacgttatc ttcttctttg aaaaccttgt cagcaacttc tggagcagaa acagcatcat 2520
aaccaccagc caaaataact ggtggggctt cttttttagc ttcaatacca tctctaccag 2580
aacctctcca agctctaaca aaaacttcat gatgtggatc tggatgagaa gtttcttcgt 2640
ccaaccatct agaattagcc aaatgcaagt aagccaaacc tggaccgtcg ttgtactctt 2700
ctctcaatct cttaatcaag aattcgaatt gtggaaccaa atcagccata acacccatac 2760
cagcatattg tgaccatgga gacaacttaa ctccaactct ttcagcacca acagcatcaa 2820
caacagctct agtaacagcc aaagcaaatc tagctctgtt ttcaatagaa ccaccccagc 2880
aatcagttct ttgattgcat ggccattgag tgaattgatc aatcaagtaa ccgttagcac 2940
catgcaattc aacaccatca aaaccaactt catgaacagc aactctagca gcggtaacga 3000
aatcagcaat gtaaccttca atttcagctt gggtcaatgg ttgtggttgt tgagattcag 3060
tttcaattgg aacatcggag gaactgacca atgggaaacc tttttctttc aacaaagcag 3120
gatcagctgc tctaccagta gcccacaatt gacagaaaat gtagcaacct cttctatgaa 3180
cagcagaaac tatacctctc caagcagcaa tttgagtagc agaatataga cctggaatat 3240
gtggttcacc agcagcttgt ggagaaatat cggtagcttc agtaatcaac aaagtacctg 3300
gaacagcagc tctttgagca taatattctt gaacgaatgg caatggagta gcagtagaat 3360
cagctctgta tctggtagtt ggagccataa tcaatctatg ttgcaagtgg cacaaaccaa 3420
tcttcatagg ttgaaacaac ttagaagctg gagcacctgg agaagatgga tcgatggtca 3480
ttttaagctt tttgtttgtt tatgtgtgtt tattcgaaac taagttcttg gtgttttaaa 3540
actaaaaaaa agactaacta taaaagtaga atttaagaag tttaagaaat agatttacag 3600
aattacaatc aatacctacc gtctttatat acttattagt caagtagggg aataatttca 3660
gggaactggt ttcaaccttt tttttcagct ttttccaaat cagagagagc agaaggtaat 3720
agaaggtgta agaaaatgag atagatacat gcgtgggtca attgccttgt gtcatcattt 3780
actccaggca ggttgcatca ctccattgag gttgtgtccg ttttttgcct gtttgtgccc 3840
ctgttctctg tagttgcgct aagagaatgg acctatgaac tgatggttgg tgaagaaaac 3900
aatattttgg tgctgggatt cttttttttt ctggatgcca gcttaaaaag cgggctccat 3960
tatatttagt ggatgccagg aataaactgt tcacccagac acctacgatg ttatatattc 4020
tgtgtaaccc gccccctatt ttgggcatgt acgggttaca gcagaattaa aaggctaatt 4080
ttttgactaa ataaagttag gaaaatcact actattaatt atttacgtat tctttgaaat 4140
ggcagtattg ataatgataa actcgaactg ggcgcgtacg cacagatatt ataacatctg 4200
cataataggc atttgcaaga attactcgtg agtaaggaaa gagtgaggaa ctatcgcata 4260
cctgcattta aagatgccga tttgggcgcg aatcctttat tttggcttca ccctcatact 4320
attatcaggg ccagaaaaag gaagtgtttc cctccttctt gaattgatgt taccctcata 4380
aagcacgtgg cctcttatcg agaaagaaat taccgtcgct cgtgatttgt ttgcaaaaag 4440
aacaaaactg aaaaaaccca gacacgctcg acttcctgtc ttcctattga ttgcagcttc 4500
caatttcgtc acacaacaag gtcctagcga cggctcacag gttttgtaac aagcaatcga 4560
aggttctgga atggcgggaa agggtttagt accacatgct atgatgccca ctgtgatctc 4620
cagagcaaag ttcgttcgat cgtactgtta ctctctctct ttcaaacaga attgtccgaa 4680
tcgtgtgaca acaacagcct gttctcacac actcttttct tctaaccaag ggggtggttt 4740
agtttagtag aacctcgtga aacttacatt tacatatata taaacttgca taaattggtc 4800
aatgcaagaa atacatattt ggtcttttct aattcgtagt ttttcaagtt cttagatgct 4860
ttctttttct cttttttaca gatcatcaag gaagtaatta tctacttttt acaacaaata 4920
taaaacaaag cttaaaatga ccatcttggt tttgggtggt agaggtaaaa ctgcttctag 4980
attgtctgct ttgttgcatg aagcgcaaat cccttttgtt gtcggtactt cttctactgc 5040
tccaattgct acttatagaa ccgctcattt tgactggttg aacaaggata cttggactaa 5100
tgcttgggat caagctccag aacctattac tgctatctat ttggttggtg ctcatgctcc 5160
agaatttgtt catccaatgt tcgaattcat cgatttggct cattctagag gtgttagaag 5220
atacgctttg gtttctgctt ctaacactga aaaaggtgat ccaatgatgg gtgaagctca 5280
tgctcatttg gattctaaag aaggtgttaa gtacgtcgtc ttaagaccaa cttggtttat 5340
ggaaaaccac atcgaagatc cacatttgtc ttggattaag accgaaaaca aaatctactc 5400
tgctactggt ccaggtaaga ttccatttgt ttctgctgat gatattgcca gagttgcttt 5460
tcatgctttg actcaaccat tgacccaaaa agaacaacaa tatatggttt tgggtccaga 5520
cttgttgtct tatgctgata tggctgaaat cttgaccgat ttgttgggta gaaagattac 5580
ccatgttact ttgccagctg aaaagttcgg tcaattcttg gttgatactg ctggtttgcc 5640
accagatttt gctgaaatgt tggctactat ggaagctgct gttgctactg gtgctgaaga 5700
aagagtttct caagacgttg aaaaggttac tggtagacca cctagaagat ttgttgatta 5760
cgctagagaa aacagagaac attggttgta accgcggatc tcttatgtct ttacgattta 5820
tagttttcat tatcaagtat gcctatatta gtatatagca tctttagatg acagtgttcg 5880
aagtttcacg aataaaagat aatattctac tttttgctcc caccgcgttt gctagcacga 5940
gtgaacacca tccctcgcct gtgagttgta cccattcctc taaactgtag acatggtagc 6000
ttcagcagtg ttcgttatgt acggcatcct ccaacaaaca gtcggttata gtttgtcctg 6060
ctcctctgaa tcgtctccct cgatatttct cattttcctt cggcgcgccg atgggacgtc 6120
agcactgtac ttgtttttgc gactagattg taaatcattc tttatttaat ctctttcttt 6180
aactactgct taaagtataa tttggtccgt agtttaataa ctatactaag cgtaacaatg 6240
catactgaca ttataagcct gaacattacg agtttaagtt gtatgtaggc gttctgtaag 6300
aggttactgc gtaaattatc aacgaatgca ttggtgtatt tgcgaaagct acttctttta 6360
acaagtattt acataagaat aatggtgatc tgctcaactg atttggtgat aactctaact 6420
tttttagcaa caatttaaaa gataattcga acatatataa cagtaggaag aatttgtgta 6480
cgtcaaatta agataattta gcattaccaa agttattaac ctaaacataa aatatatatg 6540
agacacatgt ggaaatcgta tgaaacaact gttatgaaac tgacaagaat gaatatatag 6600
agtaagctcc gcttgtaaag aggaatcact taagtgtata aatgtctcga cgattacttt 6660
agatccaaga ttgatgattg atattactct gtaatactta agctctttta atagctcact 6720
gttgtattac gggctcgagt aataccgcct gcagggcatg caagcttggc gtaatcatgg 6780
tcatagctgt ttcctgtgtg aaattgttat ccgctcacaa ttccacacaa catacgagcc 6840
ggaagcataa agtgtaaagc ctggggtgcc taatgagtga gctaactcac attaattgcg 6900
ttgcgctcac tgcccgcttt ccagtcggga aacctgtcgt gccagctgca ttaatgaatc 6960
ggccaacgcg cggggagagg cggtttgcgt attgggcgct cttccgcttc ctcgctcact 7020
gactcgctgc gctcggtcgt tcggctgcgg cgagcggtat cagctcactc aaaggcggta 7080
atacggttat ccacagaatc aggggataac gcaggaaaga acatgtgagc aaaaggccag 7140
caaaaggcca ggaaccgtaa aaaggccgcg ttgctggcgt ttttccatag gctccgcccc 7200
cctgacgagc atcacaaaaa tcgacgctca agtcagaggt ggcgaaaccc gacaggacta 7260
taaagatacc aggcgtttcc ccctggaagc tccctcgtgc gctctcctgt tccgaccctg 7320
ccgcttaccg gatacctgtc cgcctttctc ccttcgggaa gcgtggcgct ttctcatagc 7380
tcacgctgta ggtatctcag ttcggtgtag gtcgttcgct ccaagctggg ctgtgtgcac 7440
gaaccccccg ttcagcccga ccgctgcgcc ttatccggta actatcgtct tgagtccaac 7500
ccggtaagac acgacttatc gccactggca gcagccactg gtaacaggat tagcagagcg 7560
aggtatgtag gcggtgctac agagttcttg aagtggtggc ctaactacgg ctacactaga 7620
aggacagtat ttggtatctg cgctctgctg aagccagtta ccttcggaaa aagagttggt 7680
agctcttgat ccggcaaaca aaccaccgct ggtagcggtg gtttttttgt ttgcaagcag 7740
cagattacgc gcagaaaaaa aggatctcaa gaagatcctt tgatcttttc tacggggtct 7800
gacgctcagt ggaacgaaaa ctcacgttaa gggattttgg tcatgagatt atcaaaaagg 7860
atcttcacct agatcctttt aaattaaaaa tgaagtttta aatcaatcta aagtatatat 7920
gagtaaactt ggtctgacag ttaccaatgc ttaatcagtg aggcacctat ctcagcgatc 7980
tgtctatttc gttcatccat agttgcctga ctccccgtcg tgtagataac tacgatacgg 8040
gagggcttac catctggccc cagtgctgca atgataccgc gagacccacg ctcaccggct 8100
ccagatttat cagcaataaa ccagccagcc ggaagggccg agcgcagaag tggtcctgca 8160
actttatccg cctccatcca gtctattaat tgttgccggg aagctagagt aagtagttcg 8220
ccagttaata gtttgcgcaa cgttgttgcc attgctacag gcatcgtggt gtcacgctcg 8280
tcgtttggta tggcttcatt cagctccggt tcccaacgat caaggcgagt tacatgatcc 8340
cccatgttgt gcaaaaaagc ggttagctcc ttcggtcctc cgatcgttgt cagaagtaag 8400
ttggccgcag tgttatcact catggttatg gcagcactgc ataattctct tactgtcatg 8460
ccatccgtaa gatgcttttc tgtgactggt gagtactcaa ccaagtcatt ctgagaatag 8520
tgtatgcggc gaccgagttg ctcttgcccg gcgtcaatac gggataatac cgcgccacat 8580
agcagaactt taaaagtgct catcattgga aaacgttctt cggggcgaaa actctcaagg 8640
atcttaccgc tgttgagatc cagttcgatg taacccactc gtgcacccaa ctgatcttca 8700
gcatctttta ctttcaccag cgtttctggg tgagcaaaaa caggaaggca aaatgccgca 8760
aaaaagggaa taagggcgac acggaaatgt tgaatactca tactcttcct ttttcaatat 8820
tattgaagca tttatcaggg ttattgtctc atgagcggat acatatttga atgtatttag 8880
aaaaataaac aaataggggt tccgcgcaca tttccccgaa aagtgccacc tgacgtctaa 8940
gaaaccatta ttatcatgac attaacctat aaaaataggc gtatcacgag gccctttcgt 9000
ctcgcgcgtt tcggtgatga cggtgaaaac ctctgacaca tgcagctccc ggagacggtc 9060
acagcttgtc tgtaagcgga tgccgggagc agacaagccc gtcagggcgc gtcagcgggt 9120
gttggcgggt gtcggggctg gcttaactat gcggcatcag agcagattgt actgagagtg 9180
caccatatgc ggtgtgaaat accgcacaga tgcgtaagga gaaaataccg catcaggcgc 9240
cattcgccat tcaggctgcg caactgttgg gaagggcgat 9280
<210> 119
<211> 2193
<212> DNA
<213> Artificial Sequence
<220>
<223> EasE-of Aspergillus japonicus and codon optimized for S.
cerrevisiae and fused with SUMO
<400> 119
atggattaca aggatgacga cgataagtcc gactcagaag tcaatcaaga agctaagcca 60
gaggtcaagc cagaagtcaa gcctgagact cacatcaatt taaaggtgtc cgatggatct 120
tcagagatat tcttcaagat caaaaagacc actcctttaa gaaggctgat ggaagcattc 180
gctaaaagac agggtaagga aatggactcc ttaacgttct tgtacgacgg tattgaaatt 240
caagctgatc aggcccctga agatttggac atggaggata acgatattat tgaggctcac 300
agggaacaga ttggaggtca gcttaaaatg ggtcaatcca gaggtatttt gggtggtgtt 360
agacaattga tcttggttat tttggttggt gcctacttgt ctagattgtc tgctgttgat 420
gatgatagac atgattgcag atgtagacca ggtgaaccat gttggccaac tgtttctcat 480
tggtctactt tgaacgaatc tattggtggt gctttggctc atgttaagcc aattgctcat 540
gtttgtcatc aatccggtca agattcttct gcttgcgaac aagttttaca agaatccttg 600
gattctaagt ggcgtgcttc tcatactggt gctttacaag attgggtttg ggaaggtggt 660
gctgaatcta atcaaacttg ttactacttg agatcaggtc cagctggttc ttgtcatcaa 720
ggtagaattc cattatattc cgctgctgtt aagtccgctt ctgatgttca aaaggttgtt 780
gatttcacca gacaacacaa cttgagattg gttattagaa acaccggtca tgatggttct 840
ggtagatcat ctggtccaga ttctgttgaa attcataccc atcacttgaa ctccgttcaa 900
taccatccaa attttagacc agctggttcc tctgaaagac aatctgctcc aggtcaacca 960
gctgttactg ttggtgctgg tattttgttg ggtgacttgt atgctagagg tgcttctgaa 1020
ggttggatag ttgttggtgg tgaatgtcca acagttggtg ctgctggtgg ttttttacaa 1080
ggtggtggtg tttcttcttg gttgtcttat gctcatggtt tggctgttga taacgtcttg 1140
gaatacgaag ttgttactgc taagggtgaa atcgttattg ctaacgctca tcaaaaccca 1200
gatttgtttt gggctttgag aggcggtggt ggtggtactt ttggtgttgt tactcaagct 1260
acattgcaag ttcatccaga tttgccagtt tctgttgctg atgctgttgt tactggttct 1320
agagctgatg ctactttttg gtcacatggt gttgctgctt tgttgagagc tttacaattc 1380
ttgaacaacc atggtactgc cggtcaattc attttgagaa acactgatga tgataccgtc 1440
caagctagtt tgactatgta cttctctaac ttgactgttc cagctgttgc tgacgaaaga 1500
atggatcctt tgagaagagc tttggaacat aatggtcatc cataccaatt gacctctaga 1560
ttcttgccac aaatctcttc tactttcaga cataccgctg atagataccc agaagattac 1620
ggtattttga tgggttccgt tttggtttcc ttggacttgt ttaattctgc tactggtcca 1680
gctgctttgg cacaacattt tgctagattg ccaatgactc cagatgattt gttgttcact 1740
tctaacttgg gtggtagagt ctcttcattg aatagagatc cagcttctac tgctatgcat 1800
ccaggttgga gagatgctgc tcaattattg aatttcgtta gaggtgttgg tgctccatct 1860
ttggctgcta aagctactgc tttacatgaa ttgcaaaccg ttcaaatggc cagattatac 1920
gaaattgaac cagccttcca aatctcctac agaaatttgg gtgatccatc cgaaagaaga 1980
tccagagaag tttattgggg ttctaactac gctagattgg ttgaagttaa gagaagatgg 2040
gatccagaag gtttgttctt ctccaaattg ggtattgatg gtgatgcttg ggatgctgaa 2100
ggtatgtgta gacaaacaag acaaggtgct tggaatgttg ctgttaagtg ggttcaatct 2160
ttcttgggtt ctttgtctgc ttccgttgtt tga 2193
<210> 120
<211> 1020
<212> DNA
<213> Artificial Sequence
<220>
<223> EasF of A. japonicus codon optimized for S. cerrevisiae
<400> 120
atgggttcta ttaacccacc aatcttggac attagacaat ctactttcga aacctccatc 60
ccacaacaag ttactgaagg tttgactaag gatccaaaaa ctttgccagc tttgttgttc 120
tattccggtg atggtattca acattggacc agacattctt gtgctccaga tttttatcca 180
agaagagaag aaatcaacat cttgcattct gaagctgctc acatggctgc ttctatttct 240
caagattccg ttgttgttga tttgggttct gcttctttgg ataaggtctt gccattttta 300
catgctttgg aagcccaaca aaaaagagtt cattactacg ccttggactt gtcttatcca 360
gttttggctt ctaccttgtc cgaaattcca gttgctcaat tccaatacgt tcaaattgct 420
gcattgcacg gtactttcga agatggttta caatggttgc atgattcccc agaagttaga 480
gaaagaccac atcatttgtt gttgttcggt ttgaccattg gtaacttctc tagaccaaat 540
gctgctgctt tcttgagaaa cattgctgat agagctttgg ctgcttctcc agctgaatct 600
tctattttgt tgaccttgga ttcttgcaag atgccaacta aggttttgag agcttatact 660
gctgaaggtg ttgttccatt tgctttgaca tctttgtcct acgccaatca attattctgg 720
ccaggtggtg aaggtagagt ttttgatgaa gaagaatggt acttccactc cgaatggaat 780
tttgctttgg gtagacatga agctgctttg attccaagag atagagatat tactttgggt 840
ccaccattgg aacaagttgt tgttagaaga ggtgaaaagg ttagattcgg ttgctcttac 900
aaatactctg ctgctgaaag agaacatttg tttgctggtg ctggtttgga aaatgttgct 960
tcttggtctg ctaaaggttg tgatgttgct ttctaccaat tgaagatgag accagaatga 1020
<210> 121
<211> 1544
<212> DNA
<213> Artificial Sequence
<220>
<223> CA cassette including both AscI sites
<400> 121
ggcgcgccat gcagggatat cagatctcca ttaccgacat ttgggcgcta tacgtgcata 60
tgttcatgta tgtatctgta tttaaaacac ttttgtatta tttttcctca tatatgtgta 120
taggtttata cggatgattt aattattact tcaccaccct ttatttcagg ctgatatctt 180
agccttgtta ctagttagaa aaagacattt ttgctgtcag tcactgtcaa gagattcttt 240
tgctggcatt tcttctagaa gcaaaaagag cgatgcgtct tttccgctga accgttccag 300
caaaaaagac taccaacgca atatggattg tcagaatcat ataaaagaga agcaaataac 360
tccttgtctt gtatcaattg cattataata tcttcttgtt agtgcaatat catatagaag 420
tcatcgaaat agatattaag aaaaacaaac tgtacaatca atcaatcaat catcacataa 480
agcttggcct gcagggccag cttaccctta aatttatttg cactactgga aaactacctg 540
ttccatggcc aacacttgtc actactttct cttatggtgt tcaatgcttt tcccgttatc 600
cggatcatat gaaacggcat gactttttca agagtgccat gcccgaaggt tatgtacagg 660
aacgcactat atctttcaaa gatgacggga actacaagac gcgtgctgaa gtcaagtttg 720
aaggtgatac ccttgttaat cgtatcgagt taaaaggtat tgattttaaa gaagatggaa 780
acattctcgg acacaaactc gagtacaact ataactcaca caatgtatac atcacggcag 840
acaaacaaaa gaatggaatc aaagctaact tcaaaattcg ccacaacatt gaagatggat 900
ccgttcaact agcagaccat tatcaacaaa atactccaat tggcgatggc cctgtccttt 960
taccagacaa ccattacctg tcgacacaat ctgccctttc gaaagatccc aacgaaaagc 1020
gtgaccacat ggtccttctt gagtttgtaa ctgctgctgg gattacacat ggcatggatg 1080
agctctacaa ataatgaatt cgtcgaggcc gttcaggcct cgaggccgtt caggctcgac 1140
ccggggatcc gcggccgcag gcctaaattg atctagagct ttggacttct tcgccagagg 1200
tttggtcaag tctccaatca aggttgtcgg cttgtctacc ttgccagaaa tttacgaaaa 1260
gatggaaaag ggtcaaatcg ttggtagata cgttgttgac acttctaaat aagcgaattt 1320
cttatgattt atgattttta ttattaaata agttataaaa aaaataagtg tatacaaatt 1380
ttaaagtgac tcttaggttt taaaacgaaa attcttgttc ttgagtaact ctttcctgta 1440
ggtcaggttg ctttctcagg tatagcatga ggtcgctctt attgaccaca cctctaccgg 1500
catgcccatg ggttaactga tcaatgcatc ctgcatggcg cgcc 1544
<210> 122
<211> 1290
<212> DNA
<213> Artificial Sequence
<220>
<223> c-terminal truncated DmaW of A. japonicus codon optimized for S.
cerrevisiae
<400> 122
atgactgctg gtcaaggtat taagactggt aatgcttctg attgcgaagt ctacagaact 60
ttgtctgttg ctttggattt cgccaatcaa gatgaagaat tgtggtggca ttctactgct 120
ccaatgtttg ctcaaatgtt gcaatctacc aactacaact tacacgctca gtacaagcac 180
ttgttgatct acaagaagaa cgtcattcca ttcttgggtg tttacccaac taatgataag 240
ccaagatggt tgtccatttt gaccagatat ggtactccat tcgaattgtc tttgaactgt 300
tctggtccat tggttagata cacttacgaa cctattaacg ctgctactgg tactgctaga 360
gatccattca atactcatgc tgtttgggac tcattggaac aattgatggc tttacaatcc 420
ggtatcgatt tggatttgtt cagacacttc aagaacgact tgactttgtc tgctgaagaa 480
tccgaatact tgtacaagaa caacttggtc ggtgaacaaa tcagaaccca aaacaaattg 540
gctttggact tgcaagatgg tgaattcgtt gttaagactt acatctaccc agccttgaaa 600
tctttggcta ctggtagaag tatccacgaa ttggtttttg gttctgcttt cagatggtct 660
aagcaatacc cagaattgag aaagccattg gataccttgg aacaatacgt ttattctaga 720
ggtccatctt ctactgcctc tccaagatta ttgtcttgcg atttgattga ccctaccaag 780
tccagaatca aaatctactt gttggaaaga atggtcactt tggaagcctt ggaagatttg 840
tggactatgg gtggtgaaag aactgatgct tctactttgg ctggtttgga aatgattaga 900
gaattgtggg aattgatcag attgccagct ggtttacaat cttatccagc tccatatttg 960
ccaatcggta ctattccaga tgaacaattg ccattgatgg ccaactacac catccatcat 1020
gatgatccag ttccagaacc acaagtttac tttactactt tcggtagaaa cgacatgcaa 1080
attgctgatg ctttggctac tttcttcgaa cgtcgtggtt ggcacgaaat ggctagacag 1140
tacaaagctg aattgtgctc tcattaccca catgctgatc acgaaacttt gaactacttg 1200
cacgcttaca tctcattctc ttacagaaag aacaagccat acttgtccgt ctacttgcaa 1260
tctttggaaa ctggtgattg ggttacctga 1290
<210> 123
<211> 1347
<212> DNA
<213> Artificial Sequence
<220>
<223> DmaW of C. purpurea codon optimized for S. cerrevisiae
<400> 123
atgagtactg ctaaagatcc aggaaatggc gtttatgaaa ttctgtctct catttttgat 60
ttcccttcta atgaacaaag actgtggtgg cattcaacag cacctatgtt cgcagctatg 120
ttagacaacg caggttacaa tattcacgat caatacagac atttggggat attcaaaaag 180
catatcatac cttttctggg tgtttaccca actaaagaca aggaaagatg gctttctatc 240
ttgactcgat gcggattgcc attagaacta tctttgaatt gtacagattc tgtggttaga 300
tacacgtacg agccaattaa cgaagtcaca ggtaccgaaa aagacacgtt taacaccttg 360
gccatcatga cttcagtcca aaaattggca caaattcaag ccggcattga cttagaatgg 420
tttagctact ttaaagacga gttaacatta gatgaatccg aatctgcaac cctgcaatct 480
aatgaattgg taaaggaaca aatcaaaact caaaataagt tggcactaga cttgaaggag 540
tctcagttcg ccctaaaggt atacttctat ccacatctca aaagtatagc gactggtaag 600
tccacacatg atctaatctt tgattctgtg cttaagttat cccaaaagca tgattccatt 660
cagcctgcct tccaagtatt gtgtgattac gtttcaagga gaaaccattc tgcggaggtc 720
gatcaacacg gtgctctaca cgctagattg ttgtcatgcg atctcattga tccagcaaaa 780
tcaagagtta aaatctacct cttagagaaa acagtgtctt tgtcagttat ggaagatttg 840
tggacacttg gtggacaaag agtagacgct agtacaatgg atggccttga tatgttaagg 900
gagttgtggt ccctgttaaa ggttccaaca ggacatcttg aataccctaa aggctatctt 960
gaattggggg aaataccaaa cgaacagcta ccatcaatgg ctaattacac ccttcaccac 1020
aatgacccta tgccagagcc tcaagtgtac tttacagttt tcggtatgaa tgacgctgag 1080
atatcaaatg ccctgactat ctttttccag agacatggat ttgacgatat ggctaaaaac 1140
tacagagtct ttttacaaga ttcataccct taccatgatt ttgaatccct caactatcta 1200
cacgcctata tcagcttcag ctatagacgt aataagccat acttatctgt ttacctgcat 1260
acttttgaaa ctggtcgttg gccagtcttt gctgattcac caatctcttt tgatgcttat 1320
agaagatgtg aacttagtac aaaataa 1347
<210> 124
<211> 1509
<212> DNA
<213> Artificial Sequence
<220>
<223> TE cassette including both AscI sites
<400> 124
ggcgcgccat gcagggatat cagatcttag cttcaaaatg tttctactcc ttttttactc 60
ttccagattt tctcggactc cgcgcatcgc cgtaccactt caaaacaccc aagcacagca 120
tactaaattt cccctctttc ttcctctagg gtgtcgttaa ttacccgtac taaaggtttg 180
gaaaagaaaa aagagaccgc ctcgtttctt tttcttcgtc gaaaaaggca ataaaaattt 240
ttatcacgtt tctttttctt gaaaattttt ttttttgatt tttttctctt tcgatgacct 300
cccattgata tttaagttaa taaacggtct tcaatttctc aagtttcagt ttcatttttc 360
ttgttctatt acaacttttt ttacttcttg ctcattagaa agaaagcata gcaatctaat 420
ctaagtttta attacaaaaa gcttggcctg cagggccagc ttacccttaa atttatttgc 480
actactggaa aactacctgt tccatggcca acacttgtca ctactttctc ttatggtgtt 540
caatgctttt cccgttatcc ggatcatatg aaacggcatg actttttcaa gagtgccatg 600
cccgaaggtt atgtacagga acgcactata tctttcaaag atgacgggaa ctacaagacg 660
cgtgctgaag tcaagtttga aggtgatacc cttgttaatc gtatcgagtt aaaaggtatt 720
gattttaaag aagatggaaa cattctcgga cacaaactcg agtacaacta taactcacac 780
aatgtataca tcacggcaga caaacaaaag aatggaatca aagctaactt caaaattcgc 840
cacaacattg aagatggatc cgttcaacta gcagaccatt atcaacaaaa tactccaatt 900
ggcgatggcc ctgtcctttt accagacaac cattacctgt cgacacaatc tgccctttcg 960
aaagatccca acgaaaagcg tgaccacatg gtccttcttg agtttgtaac tgctgctggg 1020
attacacatg gcatggatga gctctacaaa taatgaattc gtcgaggccg ttcaggcctc 1080
gaggccgttc aggctcgacc cggggatccg cggagtgctt ttaactaaga attattagtc 1140
ttttctgctt attttttcat catagtttag aacactttat attaacgaat agtttatgaa 1200
tctatttagg tttaaaaatt gatacagttt tataagttac tttttcaaag actcgtgctg 1260
tctattgcat aatgcactgg aaggggaaaa aaaaggtgca cacgcgtggc tttttcttga 1320
atttgcagtt tgaaaaataa ctacatggat gataagaaaa catggagtac agtcactttg 1380
agaaccttca atcagctggt aacgtcttcg ttaattggat actcaaaaaa gatggatagc 1440
atgaatcaca agatggaagg aaatgcatgc ccatgggtta actgatcaat gcatcctgca 1500
tggcgcgcc 1509
<210> 125
<211> 9453
<212> DNA
<213> Artificial Sequence
<220>
<223> Fwd primer for Seq ID 126, w. homology to ERG20 prom and pEVE2049
upstream of loxP
<400> 125
ggctagctaa gatccgctct aaccgaaaag gaaggagtta gacaacctga agtctaggtc 60
cctatttatt tttttatagt tatgttagta ttaagaacgt tatttatatt tcaaattttt 120
cttttttttc tgtacagacg cgtgtacgca tgtaacatta tactgaaaac cttgcttgag 180
aaggttttgg gacgctcgaa gatccagctg cattaatgaa tcggccaacg cgcggggaga 240
ggcggtttgc gtattgggcg ctcttccgct tcctcgctca ctgactcgct gcgctcggtc 300
gttcggctgc ggcgagcggt atcagctcac tcaaaggcgg taatacggtt atccacagaa 360
tcaggggata acgcaggaaa gaacatgtga gcaaaaggcc agcaaaaggc caggaaccgt 420
aaaaaggccg cgttgctggc gtttttccat aggctccgcc cccctgacga gcatcacaaa 480
aatcgacgct caagtcagag gtggcgaaac ccgacaggac tataaagata ccaggcgttt 540
ccccctggaa gctccctcgt gcgctctcct gttccgaccc tgccgcttac cggatacctg 600
tccgcctttc tcccttcggg aagcgtggcg ctttctcata gctcacgctg taggtatctc 660
agttcggtgt aggtcgttcg ctccaagctg ggctgtgtgc acgaaccccc cgttcagccc 720
gaccgctgcg ccttatccgg taactatcgt cttgagtcca acccggtaag acacgactta 780
tcgccactgg cagcagccac tggtaacagg attagcagag cgaggtatgt aggcggtgct 840
acagagttct tgaagtggtg gcctaactac ggctacacta gaaggacagt atttggtatc 900
tgcgctctgc tgaagccagt taccttcgga aaaagagttg gtagctcttg atccggcaaa 960
caaaccaccg ctggtagcgg tggttttttt gtttgcaagc agcagattac gcgcagaaaa 1020
aaaggatctc aagaagatcc tttgatcttt tctacggggt ctgacgctca gtggaacgaa 1080
aactcacgtt aagggatttt ggtcatgaga ttatcaaaaa ggatcttcac ctagatcctt 1140
ttaaattaaa aatgaagttt taaatcaatc taaagtatat atgagtaaac ttggtctgac 1200
agttaccaat gcttaatcag tgaggcacct atctcagcga tctgtctatt tcgttcatcc 1260
atagttgcct gactccccgt cgtgtagata actacgatac gggagggctt accatctggc 1320
cccagtgctg caatgatacc gcgagaccca cgctcaccgg ctccagattt atcagcaata 1380
aaccagccag ccggaagggc cgagcgcaga agtggtcctg caactttatc cgcctccatc 1440
cagtctatta attgttgccg ggaagctaga gtaagtagtt cgccagttaa tagtttgcgc 1500
aacgttgttg ccattgctac aggcatcgtg gtgtcacgct cgtcgtttgg tatggcttca 1560
ttcagctccg gttcccaacg atcaaggcga gttacatgat cccccatgtt gtgcaaaaaa 1620
gcggttagct ccttcggtcc tccgatcgtt gtcagaagta agttggccgc agtgttatca 1680
ctcatggtta tggcagcact gcataattct cttactgtca tgccatccgt aagatgcttt 1740
tctgtgactg gtgagtactc aaccaagtca ttctgagaat agtgtatgcg gcgaccgagt 1800
tgctcttgcc cggcgtcaat acgggataat accgcgccac atagcagaac tttaaaagtg 1860
ctcatcattg gaaaacgttc ttcggggcga aaactctcaa ggatcttacc gctgttgaga 1920
tccagttcga tgtaacccac tcgtgcaccc aactgatctt cagcatcttt tactttcacc 1980
agcgtttctg ggtgagcaaa aacaggaagg caaaatgccg caaaaaaggg aataagggcg 2040
acacggaaat gttgaatact catactcttc ctttttcaat attattgaag catttatcag 2100
ggttattgtc tcatgagcgg atacatattt gaatgtattt agaaaaataa acaaataggg 2160
gttccgcgca catttccccg aaaagtgcca cctgaacgaa gcatctgtgc ttcattttgt 2220
agaacaaaaa tgcaacgcga gagcgctaat ttttcaaaca aagaatctga gctgcatttt 2280
tacagaacag aaatgcaacg cgaaagcgct attttaccaa cgaagaatct gtgcttcatt 2340
tttgtaaaac aaaaatgcaa cgcgagagcg ctaatttttc aaacaaagaa tctgagctgc 2400
atttttacag aacagaaatg caacgcgaga gcgctatttt accaacaaag aatctatact 2460
tcttttttgt tctacaaaaa tgcatcccga gagcgctatt tttctaacaa agcatcttag 2520
attacttttt ttctcctttg tgcgctctat aatgcagtct cttgataact ttttgcactg 2580
taggtccgtt aaggttagaa gaaggctact ttggtgtcta ttttctcttc cataaaaaaa 2640
gcctgactcc acttcccgcg tttactgatt actagcgaag ctgcgggtgc attttttcaa 2700
gataaaggca tccccgatta tattctatac cgatgtggat tgcgcatact ttgtgaacag 2760
aaagtgatag cgttgatgat tcttcattgg tcagaaaatt atgaacggtt tcttctattt 2820
tgtctctata tactacgtat aggaaatgtt tacattttcg tattgttttc gattcactct 2880
atgaatagtt cttactacaa tttttttgtc taaagagtaa tactagagat aaacataaaa 2940
aatgtagagg tcgagtttag atgcaagttc aaggagcgaa aggtggatgg gtaggttata 3000
tagggatata gcacagagat atatagcaaa gagatacttt tgagcaatgt ttgtggaagc 3060
ggtattcgca atattttagt agctcgttac agtccggtgc gtttttggtt ttttgaaagt 3120
gcgtcttcag agcgcttttg gttttcaaaa gcgctctgaa gttcctatac tttctagaga 3180
ataggaactt cggaatagga acttcaaagc gtttccgaaa acgagcgctt ccgaaaatgc 3240
aacgcgagct gcgcacatac agctcactgt tcacgtcgca cctatatctg cgtgttgcct 3300
gtatatatat atacatgaga agaacggcat agtgcgtgtt tatgcttaaa tgcgtactta 3360
tatgcgtcta tttatgtagg atgaaaggta gtctagtacc tcctgtgata ttatcccatt 3420
ccatgcgggg tatcgtatgc ttccttcagc actacccttt agctgttcta tatgctgcca 3480
ctcctcaatt ggattagtct catccttcaa tgctatcatt tcctttgata ttggatcata 3540
ctaagaaacc attattatca tgacattaac ctataaaaat aggcgtatca cgaggccctt 3600
tcgtctcgcg cgtttcggtg atgacggtga aaacctctga cacatgcagc tcccggagac 3660
ggtcacagct tgtctgtaag cggatgccgg gagcagacaa gcccgtcagg gcgcgtcagc 3720
gggtgttggc gggtgtcggg gctggcttaa ctatgcggca tcagagcaga ttgtactgag 3780
agtgcaccat accacagctt ttcaattcaa ttcatcattt tttttttatt cttttttttg 3840
atttcggttt ctttgaaatt tttttgattc ggtaatctcc gaacagaagg aagaacgaag 3900
gaaggagcac agacttagat tggtatatat acgcatatgt agtgttgaag aaacatgaaa 3960
ttgcccagta ttcttaaccc aactgcacag aacaaaaacc tgcaggaaac gaagataaat 4020
catgtcgaaa gctacatata aggaacgtgc tgctactcat cctagtcctg ttgctgccaa 4080
gctatttaat atcatgcacg aaaagcaaac aaacttgtgt gcttcattgg atgttcgtac 4140
caccaaggaa ttactggagt tagttgaagc attaggtccc aaaatttgtt tactaaaaac 4200
acatgtggat atcttgactg atttttccat ggagggcaca gttaagccgc taaaggcatt 4260
atccgccaag tacaattttt tactcttcga agacagaaaa tttgctgaca ttggtaatac 4320
agtcaaattg cagtactctg cgggtgtata cagaatagca gaatgggcag acattacgaa 4380
tgcacacggt gtggtgggcc caggtattgt tagcggtttg aagcaggcgg cagaagaagt 4440
aacaaaggaa cctagaggcc ttttgatgtt agcagaattg tcatgcaagg gctccctatc 4500
tactggagaa tatactaagg gtactgttga cattgcgaag agcgacaaag attttgttat 4560
cggctttatt gctcaaagag acatgggtgg aagagatgaa ggttacgatt ggttgattat 4620
gacacccggt gtgggtttag atgacaaggg agacgcattg ggtcaacagt atagaaccgt 4680
ggatgatgtg gtctctacag gatctgacat tattattgtt ggaagaggac tatttgcaaa 4740
gggaagggat gctaaggtag agggtgaacg ttacagaaaa gcaggctggg aagcatattt 4800
gagaagatgc ggccagcaaa actaaaaaac tgtattataa gtaaatgcat gtatactaaa 4860
ctcacaaatt agagcttcaa tttaattata tcagttatta ccctatgcgg tgtgaaatac 4920
cgcacagatg cgtaaggaga aaataccgca tcaggaaatt gtaaacgtta atattttgtt 4980
aaaattcgcg ttaaattttt gttaaatcag ctcatttttt aaccaatagg ccgaaatcgg 5040
caaaatccct tataaatcaa aagaatagac cgagataggg ttgagtgttg ttccagtttg 5100
gaacaagagt ccactattaa agaacgtgga ctccaacgtc aaagggcgaa aaaccgtcta 5160
tcagggcgat ggcccactac gtgaaccatc accctaatca agttttttgg ggtcgaggtg 5220
ccgtaaagca ctaaatcgga accctaaagg gagcccccga tttagagctt gacggggaaa 5280
gccggcgaac gtggcgagaa aggaagggaa gaaagcgaaa ggagcgggcg ctagggcgct 5340
ggcaagtgta gcggtcacgc tgcgcgtaac caccacaccc gccgcgctta atgcgccgct 5400
acagggcgcg tccattcgcc attcaggctg cgcaactgtt gggaagggcg atcggtgcgg 5460
gcctcttcgc tattacgcca gctgaattgg agcgacctca tgctatacct gagaaagcaa 5520
cctgacctac aggaaagagt tactcaagaa taagaatttt cgttttaaaa cctaagagtc 5580
actttaaaat ttgtatacac ttattttttt tataacttat ttaataataa aaatcataaa 5640
tcataagaaa ttcgcttatt tagaagtgtc aacaacgtat ctaccaacga tttgaccctt 5700
ttccatcttt tcgtaaattt ctggcaaggt agacaagccg acaaccttga ttggagactt 5760
gaccaaacct ctggcgaaga attgttaatt aagagctcgt ttggctagta ctgggtgatg 5820
acgccaggct ctgatccatc tggggttagt ggtagttttg tctccctaac ggtggttccg 5880
gcggtcttgg tcagcttgga cagctcttgg acggtggctg aaaggccagg gtccacatgg 5940
acagagttga aggatgaggt aacccaatca ccagtttcca aagattgcaa gtagacggac 6000
aagtatggct tgttctttct gtaagagaat gagatgtaag cgtgcaagta gttcaaagtt 6060
tcgtgatcag catgtgggta atgagagcac aattcagctt tgtactgtct agccatttcg 6120
tgccaaccac gacgttcgaa gaaagtagcc aaagcatcag caatttgcat gtcgtttcta 6180
ccgaaagtag taaagtaaac ttgtggttct ggaactggat catcatgatg gatggtgtag 6240
ttggccatca atggcaattg ttcatctgga atagtaccga ttggcaaata tggagctgga 6300
taagattgta aaccagctgg caatctgatc aattcccaca attctctaat catttccaaa 6360
ccagccaaag tagaagcatc agttctttca ccacccatag tccacaaatc ttccaaggct 6420
tccaaagtga ccattctttc caacaagtag attttgattc tggacttggt agggtcaatc 6480
aaatcgcaag acaataatct tggagaggca gtagaagatg gacctctaga ataaacgtat 6540
tgttccaagg tatccaatgg ctttctcaat tctgggtatt gcttagacca tctgaaagca 6600
gaaccaaaaa ccaattcgtg gatacttcta ccagtagcca aagatttcaa ggctgggtag 6660
atgtaagtct taacaacgaa ttcaccatct tgcaagtcca aagccaattt gttttgggtt 6720
ctgatttgtt caccgaccaa gttgttcttg tacaagtatt cggattcttc agcagacaaa 6780
gtcaagtcgt tcttgaagtg tctgaacaaa tccaaatcga taccggattg taaagccatc 6840
aattgttcca atgagtccca aacagcatga gtattgaatg gatctctagc agtaccagta 6900
gcagcgttaa taggttcgta agtgtatcta accaatggac cagaacagtt caaagacaat 6960
tcgaatggag taccatatct ggtcaaaatg gacaaccatc ttggcttatc attagttggg 7020
taaacaccca agaatggaat gacgttcttc ttgtagatca acaagtgctt gtactgagcg 7080
tgtaagttgt agttggtaga ttgcaacatt tgagcaaaca ttggagcagt agaatgccac 7140
cacaattctt catcttgatt ggcgaaatcc aaagcaacag acaaagttct gtagacttcg 7200
caatcagaag cattaccagt cttaatacct tgaccagcag tcattttaag ctttttgtaa 7260
ttaaaactta gattagattg ctatgctttc tttctaatga gcaagaagta aaaaaagttg 7320
taatagaaca agaaaaatga aactgaaact tgagaaattg aagaccgttt attaacttaa 7380
atatcaatgg gaggtcatcg aaagagaaaa aaatcaaaaa aaaaaatttt caagaaaaag 7440
aaacgtgata aaaattttta ttgccttttt cgacgaagaa aaagaaacga ggcggtctct 7500
tttttctttt ccaaaccttt agtacgggta attaacgaca ccctagagga agaaagaggg 7560
gaaatttagt atgctgtgct tgggtgtttt gaagtggtac ggcgatgcgc ggagtccgag 7620
aaaatctgga agagtaaaaa aggagtagaa acattttgaa gctaagatct acgcacagat 7680
attataacat ctgcataata ggcatttgca agaattactc gtgagtaagg aaagagtgag 7740
gaactatcgc atacctgcat ttaaagatgc cgatttgggc gcgaatcctt tattttggct 7800
tcaccctcat actattatca gggccagaaa aaggaagtgt ttccctcctt cttgaattga 7860
tgttaccctc ataaagcacg tggcctctta tcgagaaaga aattaccgtc gctcgtgatt 7920
tgtttgcaaa aagaacaaaa ctgaaaaaac ccagacacgc tcgacttcct gtcttcctat 7980
tgattgcagc ttccaatttc gtcacacaac aaggtcctag cgacggctca caggttttgt 8040
aacaagcaat cgaaggttct ggaatggcgg gaaagggttt agtaccacat gctatgatgc 8100
ccactgtgat ctccagagca aagttcgttc gatcgtactg ttactctctc tctttcaaac 8160
agaattgtcc gaatcgtgtg acaacaacag cctgttctca cacactcttt tcttctaacc 8220
aagggggtgg tttagtttag tagaacctcg tgaaacttac atttacatat atataaactt 8280
gcataaattg gtcaatgcaa gaaatacata tttggtcttt tctaattcgt agtttttcaa 8340
gttcttagat gctttctttt tctctttttt acagatcatc aaggaagtaa ttatctactt 8400
tttacaacaa atataaaaca aagcttaaaa tgacaatttc tgctccacct attattgata 8460
ttagacaagc agggttggaa agttctatac cagaccaagt tgtcgaaggt ctaactaagg 8520
aagtcaagac acttcctgca cttttgtttt actcaacaaa aggcattcag cattggaata 8580
gacattccca cgctgctgat ttctatccta gacatgagga actatgcatc ctcaaagctg 8640
aagctagtaa gatggctgct tcaatcgctc aagatagctt agtaatcgat atgggttctg 8700
ccagtatgga taaggttatc ctcctgttgg aagcgttaga ggaacagaaa aagtccataa 8760
cttactacgc cttggacctg tcctactctg aattagcatc taattttcaa gccattccag 8820
tggacagatt ccactatgtt agattcgcag ccttacatgg cacttttgac gatggacttc 8880
actggttaca aaacgctcct gatattagga acagaccaag atgtatactg ttatttgggt 8940
taaccatcgg taacttctca cgagataatg ctgcatcttt cctcagaaat atcgcccaat 9000
ctgctctgtc tacatcccca actcaatcat ccattatagt gtctctagat tcatgtaaac 9060
ttcctacgaa aatcttaaga gcctacaccg cagatggagt agttccattt gctcttgcct 9120
cactaagcta tgcgaattca ttgtttcatc caaaaggcga cagaaagatt ttcaatgaag 9180
aggattggta ctttcattct gaatggaacc atgcattggg tcgtcacgaa gctagtttga 9240
tcacacaatc aaaagatatc caattaggtg caccactaga gactgttata gtcagacgtg 9300
atgaaaagat cagatttgga tgctcttaca aatacgacaa agctgaaagg gatcagttgt 9360
ttcattcagc gggtttggag gatgccgcag tgtggacagc accagactgt gacgtagcat 9420
tctatcaatt gagactaaga ttgaactaac cgc 9453
<210> 126
<211> 4762
<212> DNA
<213> Artificial Sequence
<220>
<223> pEVE2049_LoxP-NatR-LoxP-Kex2 promoter
<400> 126
tcgcgcgttt cggtgatgac ggtgaaaacc tctgacacat gcagctcccg gagacggtca 60
cagcttgtct gtaagcggat gccgggagca gacaagcccg tcagggcgcg tcagcgggtg 120
ttggcgggtg tcggggctgg cttaactatg cggcatcaga gcagattgta ctgagagtgc 180
accatatgcg gtgtgaaata ccgcacagat gcgtaaggag aaaataccgc atcaggcgcc 240
attcgccatt caggctgcgc aactgttggg aagggcgatc ggtgcgggcc tcttcgctat 300
tacgccagct ggcgaaaggg ggatgtgctg caaggcgatt aagttgggta acgccagggt 360
tttcccagtc acgacgttgt aaaacgacgg ccagtgaatt cgagctccct gcaggatcgt 420
caaggttcgt attgttggtt tagccggtgt gcattttaag aaaatttctg ggaacacttg 480
gttgtataaa gagctggctt ctagtatatt aaaagaacta aagttgtaag taacacgttc 540
cctgagaaac cacagcttac atttttgata ttcgaaaaca agaatatccg ttcgatagaa 600
atttatatat atatatagat tctttttatt ctcttttgga aagaagaaat agaatggacc 660
taagcagaca ctcagcagtt aatattttcg tcccttaatt ggtattattt ataaataaat 720
ggcacgtact ttatgtgtaa agcagttcat catcgattga tggaggcggc cgcctaataa 780
cttcgtatag catacattat acgaagttag gtcacccggc cagcgacatg gaggcccaga 840
ataccctcct tgacagtctt gacgtgcgca gctcaggggc atgatgtgac tgtcgcccgt 900
acatttagcc catacatccc catgtataat catttgcatc catacatttt gatggccgca 960
cggcgcgaac gaaaaattac ggctcctcgc tgcagacctg cgagcaggga aacgctcccc 1020
tcacagacgc gttgaattgt ccccacgccg cgcccctgta gagaaatata aaaggttagg 1080
atttgccact gaggttcttc tttcatatac ttccttttaa aatcttgcta ggatacagtt 1140
ctcacatcac atccgaacat aaacaaccat gggtaccact cttgacgaca cggcttaccg 1200
gtaccgcacc agtgtcccgg gggacgccga ggccatcgag gcactggatg ggtccttcac 1260
caccgacacc gtcttccgcg tcaccgccac cggggacggc ttcaccctgc gggaggtgcc 1320
ggtggacccg cccctgacca aggtgttccc cgacgacgaa tcggacgacg aatcggacgc 1380
cggggaggac ggcgacccgg actcccggac gttcgtcgcg tacggggacg acggcgacct 1440
ggcgggcttc gtggtcgtct cgtactccgg ctggaaccgc cggctgaccg tcgaggacat 1500
cgaggtcgcc ccggagcacc gggggcacgg ggtcgggcgc gcgttgatgg ggctcgcgac 1560
ggagttcgcc cgcgagcggg gcgccgggca cctctggctg gaggtcacca acgtcaacgc 1620
accggcgatc cacgcgtacc ggcggatggg gttcaccctc tgcggcctgg acaccgccct 1680
gtacgacggc accgcctcgg acggcgagca ggcgctctac atgagcatgc cctgccccta 1740
atcagtactg acaataaaaa gattcttgtt ttcaagaact tgtcatttgt atagtttttt 1800
tatattgtag ttgttctatt ttaatcaaat gttagcgtga tttatatttt ttttgcctcg 1860
acatcatctg cccagatgcg aagttaagtg cgcagaaagt aatatcatgc gtcaatcgta 1920
tgtgaatgct ggtcgctata ctgctgtcga ttcgatacta acgccgccat ccagtgtcga 1980
aataacttcg tatagcatac attatacgaa gttagcggcc gccctaggcc gcggtcagca 2040
gctctgatgt agatacacgt atctcgacat gttttatttt tactatacat acataaaaga 2100
aataaaaaat gataacgtgt atattattat tcatataatc aatgagggtc attttctgaa 2160
acgcaaaaaa cggtaaatgg aaaaaaaata aagatagaaa aagaaaacaa acaaaggaaa 2220
ggttagcata ttaaataact gagctgatac ttcaacagca tcgctgaaga gaacagtatt 2280
gaaaccgaaa cattttctaa aggcaaacaa ggtactccat atttgctgga cgtgttcttt 2340
ctctcgtttc atatgcataa ttctgtcata agcctgttct ttttcctggc ttaaacatcc 2400
cgttttgtaa aagagaaatc tattccacat atttcattca ttcggctacc atactaagga 2460
taaactaatc ccgttgtttt ttggcctcgt cacataatta taaactacta acccattatc 2520
agaagcttgg cgtaatcatg gtcatagctg tttcctgtgt gaaattgtta tccgctcaca 2580
attccacaca acatacgagc cggaagcata aagtgtaaag cctggggtgc ctaatgagtg 2640
agctaactca cattaattgc gttgcgctca ctgcccgctt tccagtcggg aaacctgtcg 2700
tgccagctgc attaatgaat cggccaacgc gcggggagag gcggtttgcg tattgggcgc 2760
tcttccgctt cctcgctcac tgactcgctg cgctcggtcg ttcggctgcg gcgagcggta 2820
tcagctcact caaaggcggt aatacggtta tccacagaat caggggataa cgcaggaaag 2880
aacatgtgag caaaaggcca gcaaaaggcc aggaaccgta aaaaggccgc gttgctggcg 2940
tttttccata ggctccgccc ccctgacgag catcacaaaa atcgacgctc aagtcagagg 3000
tggcgaaacc cgacaggact ataaagatac caggcgtttc cccctggaag ctccctcgtg 3060
cgctctcctg ttccgaccct gccgcttacc ggatacctgt ccgcctttct cccttcggga 3120
agcgtggcgc tttctcatag ctcacgctgt aggtatctca gttcggtgta ggtcgttcgc 3180
tccaagctgg gctgtgtgca cgaacccccc gttcagcccg accgctgcgc cttatccggt 3240
aactatcgtc ttgagtccaa cccggtaaga cacgacttat cgccactggc agcagccact 3300
ggtaacagga ttagcagagc gaggtatgta ggcggtgcta cagagttctt gaagtggtgg 3360
cctaactacg gctacactag aaggacagta tttggtatct gcgctctgct gaagccagtt 3420
accttcggaa aaagagttgg tagctcttga tccggcaaac aaaccaccgc tggtagcggt 3480
ggtttttttg tttgcaagca gcagattacg cgcagaaaaa aaggatctca agaagatcct 3540
ttgatctttt ctacggggtc tgacgctcag tggaacgaaa actcacgtta agggattttg 3600
gtcatgagat tatcaaaaag gatcttcacc tagatccttt taaattaaaa atgaagtttt 3660
aaatcaatct aaagtatata tgagtaaact tggtctgaca gttaccaatg cttaatcagt 3720
gaggcaccta tctcagcgat ctgtctattt cgttcatcca tagttgcctg actccccgtc 3780
gtgtagataa ctacgatacg ggagggctta ccatctggcc ccagtgctgc aatgataccg 3840
cgagacccac gctcaccggc tccagattta tcagcaataa accagccagc cggaagggcc 3900
gagcgcagaa gtggtcctgc aactttatcc gcctccatcc agtctattaa ttgttgccgg 3960
gaagctagag taagtagttc gccagttaat agtttgcgca acgttgttgc cattgctaca 4020
ggcatcgtgg tgtcacgctc gtcgtttggt atggcttcat tcagctccgg ttcccaacga 4080
tcaaggcgag ttacatgatc ccccatgttg tgcaaaaaag cggttagctc cttcggtcct 4140
ccgatcgttg tcagaagtaa gttggccgca gtgttatcac tcatggttat ggcagcactg 4200
cataattctc ttactgtcat gccatccgta agatgctttt ctgtgactgg tgagtactca 4260
accaagtcat tctgagaata gtgtatgcgg cgaccgagtt gctcttgccc ggcgtcaata 4320
cgggataata ccgcgccaca tagcagaact ttaaaagtgc tcatcattgg aaaacgttct 4380
tcggggcgaa aactctcaag gatcttaccg ctgttgagat ccagttcgat gtaacccact 4440
cgtgcaccca actgatcttc agcatctttt actttcacca gcgtttctgg gtgagcaaaa 4500
acaggaaggc aaaatgccgc aaaaaaggga ataagggcga cacggaaatg ttgaatactc 4560
atactcttcc tttttcaata ttattgaagc atttatcagg gttattgtct catgagcgga 4620
tacatatttg aatgtattta gaaaaataaa caaatagggg ttccgcgcac atttccccga 4680
aaagtgccac ctgacgtcta agaaaccatt attatcatga cattaaccta taaaaatagg 4740
cgtatcacga ggccctttcg tc 4762
<210> 127
<211> 68
<212> DNA
<213> Artificial Sequence
<220>
<223> Fwd primer for Seq ID 126, w. homology to ERG20 prom and pEVE2049
upstream of loxP
<400> 127
cattattctt aaatgttccg atcctcttgt tcgcatatat acagtcgatt gatggaggcg 60
gccgccta 68
<210> 128
<211> 73
<212> DNA
<213> Artificial Sequence
<220>
<223> Rev primer for Seq ID 126, w homology for Kex2 promoter and 5-end
of ERG20 ORF
<400> 128
ctactaaccc attatcagaa gacaatggct tcagaaaaag aaattaggag agagagattc 60
ttgaacgttt tcc 73
<210> 129
<211> 1326
<212> DNA
<213> Paecilomyces divaricatus
<400> 129
atgagttcca gaaagactcg aaatgcgtct agttcagaag tctaccagac tctcagcagt 60
ctcttcgact tccctgacga ggagcagaag ctctggtggc acagtactgc gcccatgttt 120
gccgacatgc tccagacggc caactacgac acccatgccc agtatcgaca tctggggatt 180
tacaagaaat acgtgatccc gttcctaggt gtctacccca ctaatgaccg tgatcgatgg 240
ctgagcattc tgacgcgcta cggtacaccg tttgagttga gtctcaactg ttcggatgac 300
gttgtgcggt acacattcga gcccatcaac gctgcgacgg gcaccatcca ggaccccttc 360
aacactcacg ccatctggga ctgtctgcag aacttcagcc caatgctgaa aggggtggac 420
ctcgaatggt ttagctattt caaagagaaa ctcaccctca atgcggacga atctgccttc 480
cttgtggaga ataacttggc tgatgatccg atcaagaccc agaacaagct ggccctggat 540
cttaagaaca gcggctttgt gctgaagacg tacatctatc cggccctcaa gtccatcgcc 600
acgggccttg cggctccctt gtcggtgata gatgagtatg tccactcccg ggggcctaat 660
ggcacggccg gccctcgcct cttctcctgt gacttggttc atccgagcaa gtctcgcatc 720
aagatctata ttctggagcg gatggtctcg ttagccgcca tggaggatct ctggacgcta 780
ggtggccgac gcaaggatca ctcgactctg gcaggattga atatgatccg tgagctctgg 840
gagctgatcc aactgcctgc cgggctgcgt tcctacccgg agccatacct gcctctgggt 900
accattccga atgagcagct gccgttgatg gccaattaca cgcttcacca ggacgatccc 960
tggcccgagc cccaagtgta cttcacccca tttggcatga acgacatggc gattgccgat 1020
gcattgacga cgttcatgga gcgccggggg tggaacgaca tggctcagtc gtacaaggac 1080
aatttgtgct catattatcc caacgcagac cacgatcatc tgaactacgt gcacgcatat 1140
atatccttct cgtaccggaa tgacaagccg tatttgagcg tgtacctaca ctccttcgag 1200
acgggggatt ggaagatctc gaacttcacc aatcccgaat cgaagtgcaa agaaccacag 1260
cgtgtgtctg tccgatttcc tgtacaagga agaggaagag gtcggatccc tgcaagtgga 1320
tattga 1326
<210> 130
<211> 1140
<212> DNA
<213> Paecilomyces divaricatus
<400> 130
atgacacaag cttcacctgc atctaagctc ttcacgccgt taagcgtcgg ttcatgcaaa 60
ctataccaca gactcgtcat ggcgccgatg actcgatttc gagcagatga gtctgccatc 120
cagctacctt ttgtcaaaga atactatgga cagcgggcat ctgtccccgg aacattgctg 180
ataagcgagg caaccgacat atctccccaa gctgggggat tcgcgaatgt cccgggtatc 240
tggaatgaag cgcagattca agcatggagg cagattgtag gcgaggtgca ttccaagggc 300
tcctttatct tttgccagct gtgggcgacg ggtcgcgctg ctgaacccag tgtgctcgcc 360
gagaagggat tcacgctagt ttccagcagt gcgaccccaa tatcgcccga cggactgtta 420
ccatgtgcac tgactgaggt cgaaatccac gattatatcg atgacttcgc acaagcagcg 480
cagaacgcaa tactcgccgg atttgatggt gttgagattc acggcgcaaa cggctatctt 540
attgaccagt tcacgcaagc ttcatgcaac aagcggattg atcactgggg tggaagtatt 600
gcgaaccgtg cccgattcgc gatcgaagtg actagggccg tcgttgctgc cgttggtgca 660
gatcgagtgg gtgtgaagct atctccatgg agtcagtacc agggcatggg gaatatggac 720
ggtctagtgg cgcaatttga gtatctcatc tcccaactgc agaagatgga tcttgcatac 780
ctgcatctgg ctaattcaac atggctggga gaggagggtc ctcattcaga ccctcaccat 840
gaagtgtttg tgcaggcgtg gggaaagtcc aaacccatct tgttggcagg agggtatgat 900
gccgcgtcgg caaaacaggt tgtggatatg ctttatggcg aacacgacaa tgttgcaatc 960
gcgttcgggc ggtttttcat ttcgaaccct gatcttccct ttcgggtaaa gtcgaatgtc 1020
aagcttcaag actatagtcg ttcttcgatg gataccagcg tgtcgaaaga cggatatttg 1080
gatcatccat tcagtccgga gtttctggct tccaacagat ctgaatttgc cagtacttag 1140
<210> 131
<211> 1560
<212> DNA
<213> Paecilomyces divaricatus
<400> 131
atgagaacaa aggacgacaa cgctcccaag ccggaccccg atggggtgcc cgcgtacacg 60
acttcaaatg gctgtcccgt catgaggccc gagggactgc cagcagacgg caggaagctc 120
ctcctaaacg actcccacct cattgacatg ttagcccact tcaacagaga gaaaatacct 180
gagcgtgcag tccatgccaa aggcgccgga gcatacggag agttcgaggt caccgaggat 240
atctctgaca tttgcaacat cgacatgttg ctaggagtcg gcaagaagac tccctgtgtc 300
acgcgctttt ccaccaccgg gctggagcgc gggtcgtctg atggcgtccg agacctcaag 360
ggtctggcaa cgaagttcta caccaagcag ggcaactggg attgggtctg cctcaacttt 420
cccttcttct tcatccggga tccgatgaag ttccccagct tgatgcacgc ccagcgccgg 480
gatccccaga ccaacctgct gaacccaaat atgtactggg attgggtgac gagcaaccat 540
gaatctctgc acatggtgct gctgcaattc agtgacttcg gcaccatgtt caactggcgc 600
tcaatgagcg gatacatagg tcacgcatac aaatgggtca tgccagacgg ctcctacaaa 660
tatgtgcaca tcttcctttc gtcaaaccag ggccccaatt tcacggacgg cggccccgac 720
aaaaaagaca atgggctgga accagaccat gccaccaggg acctttacga agcgatcgag 780
agcggtgatt acccgagctg gacggccaat gtgcaagtag tcgacccgaa agatgctccg 840
aagctgggat ttaacattct cgatatcacc aagcactgga atctcggcac atacccgaag 900
gatctggaag ccattccgtc cagaccgttc ggaaagctga ctctgaaccg caacccacaa 960
aactatttcg ctgaaatcga gcagctcgca ttttccccgt ctaatatggt ccccggtgtc 1020
gagccatcgg aggatccaat cctccaggcg cgcatgtttg cctatccgga tgctcagcgc 1080
taccgcctgg gcgtgaacaa ccaacatctg cccgtcaatc gaccccagca cgtcttcaat 1140
ccccttctgc gagacggtgc tggaatggcc tctggcaact acggctccct gcccggctac 1200
gtgtccgact atcacccaat gaagtttcag aaaccaacac cccagaaccc ggagttcgac 1260
tcgtggctgg ctcagatctc ctcgcagtca tggggccaat caaatgagca ggactacaag 1320
ttcgccaggg aatactactt ggcactgccg gagttccgaa gccaggagtt ccaggacact 1380
atggtccgaa atatttgtca atctgtggca cagacgaatc gcgagatacg cgagaagaca 1440
taccgcacgt tcgacctggt ccaccggggg ctggcggatc gtatccggca gggggtggag 1500
gagctggttg cggaggcgtt cgtcatccca cgaggatcat cttcgtcact gagattatag 1560
<210> 132
<211> 786
<212> DNA
<213> Paecilomyces divaricatus
<400> 132
atggcgtctg tatcatctag gatctttgcc atcactggcg gcgcatcagg catcggtgcc 60
gcaacttgtc gcctactcgc ccaacgcggc gcagccgtca tctgcgttgg cgacatctca 120
catgggaact ttgacgatct gaaaaaatcg attaaagaga tcaacccatc aacagaggtt 180
cactgcacgt ctctggatgt gacctcagtt gctgaggtag acaaatggat acagagtatc 240
atctcaagct ttggggactt acatggagct gcaaatgttg ctggcatcgc ccagggtgca 300
ggcatgcgaa agtccccaac cattgtggag gagacagacg aagaatggaa taagattttc 360
aaagtcaatc tagacggtgt tttctactgc actcgggctg aggtgcgggc tatgaaggac 420
cttccccgag gaagtcgcag cattgtcaat gttggcagca ttgctgctct ctcccatatg 480
ccggatgtct atgcgtacgg gacgtcgaaa ggggctgcgg cctacttcac tacgtgtgcg 540
gcggcggatt cgtttccact agggattcgg attaatggtg tttctccagg aatcacggac 600
acccagctac taccgcagtt catgccgggc gcaaaatcgg ccgatgaagt gaaagaagca 660
tacgagaagg agggtttcac aatcatcaag ccggatgatg tcgcacggac gatcgtatgg 720
ttgctgagtg aagattcaac tcccgtgtat ggggccaaca tcaacgtcgg tgctggcgtg 780
ccctag 786
<210> 133
<211> 1830
<212> DNA
<213> Paecilomyces divaricatus
<400> 133
atgcgatacg ttctcctcgc cgtggcaggt cttctcttgg gctggctctc ctttacgggg 60
aaggattccc ggccatgtcg ttgtagaccg tggacatcat gctggccctc ggaagagacg 120
tggagggcct tcaacaaatc catagatggc aatctaatcc gtctcaggcc catagcccat 180
gtgtgccatg agcccacgtt caaccagagc gcctgtaacg aactgcgtcg tttttcactg 240
gacagcggat ggagggctgc acaaccaggg gcattgcagg actgggtctg ggagagtggc 300
tccaaccctg acgaggcctg tcttatggca tcaccgcgcg agatgccctg tcaccaaggc 360
cgagtccctc tcttttcggc tgctgttgac tctgctgagc aggtccagcg ggcggttcga 420
ttcgctaaac ggtacaacct ccgtctcgtc attcgcaata cgggccatga cggtgcggga 480
cagtccagtg gtcccgattc gttccagatc catacccatc gcctgcaggc aattcagtac 540
cacacggact ttcagccaaa tgggtctctc gtacgtctgg gctctgccgt gactgtggga 600
gccggggtgc agctcgggga cctgtacgca cgaggctcgc tcgagggata tactgtggtc 660
gggggtgaat gtccaaccgt gggtgccgct ggcggtttcc ttcagggcgg aggtgtatct 720
agtttcctga gtcacagctg ggggctggct gtggataatg ttctcgaatt cgagatggtg 780
accgcccagg gagacctggt cgtcgccaac gcacaccaaa accaagacct tttctgggcg 840
ttgagaggag gcggaggcgg cacctttggt gtcgtgactc gagccactct tcgagcgtat 900
catgatctcc ccgttgtagt ggcgaagctc accatcgcag ccctcgacgc ggatgcaatc 960
ttctggacca aaggcgtcac cactcttctc acagcactgc agaaattcaa ccgtgaaagt 1020
gttgccggcc aattcctctt aaaaccccag acggagagcg gaattgaagc cagcattacg 1080
atgtatttca tcaacgagac tgagacggcg cgtgttgacg accgcctaaa tccgctgctg 1140
accatcctta acagcgacgg catcacatct gctcggtcat cgcaattcct gccccggctc 1200
agcatggatc tccgaacagc acccgacatc tatcctgaga actacgggat tctaatggcg 1260
tctgtcctcg tttcgaacca gctgttcaat tggtccgagg gacctcaacg aatggcggag 1320
aactttgcac gactacctat gggaccccat gatctgctct tcaccagcaa ccttggcggc 1380
cgtgtcatag ccaacggaga cgtcgtggac acggcgatgc atcctgcctg gcgctctgcc 1440
gcccagctca tcaacttggt tcgggccgtt gatccgtcca tatcaggcaa gtccacggct 1500
ttagaaggcc ttacgaacat ccaaatgcca atcctatatt ctctcgatcc ggcgtttagg 1560
gcctcttatc tgaatctggc ggatcccaat gagaaagact ctccccaggt cttctgggga 1620
gagaactatg gacgtctgtt acagatcaag cggaagtggg atgcaggtga tctcttcctc 1680
acgaagctgg gagtgggtag cgatgactgg gagatggacg ggatgtgtag aaagcggcag 1740
aatctgataa atcgtgcgtt taagtctgtt ccaagccaag ttggcaagac cctgtggcgc 1800
ttgctggagg gcagttcgaa tgatttgtaa 1830
<210> 134
<211> 1026
<212> DNA
<213> Paecilomyces divaricatus
<400> 134
atgccgacct ctcgaatcat cgatatccgc cgttccaaat tcgaaggttc gattccggac 60
caggtcgtcg acgggctcac gagcagcccc aagaccctcc cggctctcct cttctacagt 120
acagagggaa tccagcactg gaatcgccac tcccactctc cggatttcta cccgagactt 180
gaggagatcc ggattctgaa gaaacaggcc tctgtgatag ccacatccat tgccgataac 240
agcgttgtcg ttgatctggg cagcgccacc ctggacaaag tgaccttcct cctggaagca 300
cttgaagccc agcagaaaaa catagactac tatgcattgg acttgagccc ctcggagctg 360
gctgcaacgc tcgaggccgt gccaatccat caattccacc acgtcaggtt cgctggcctc 420
catggcacct ttgacgacgg cttgcaatgg ctgaaggaga caccggagat ctgcgaccgg 480
ccgcattgcg ttctcctgtt agggctgacg attggcaact tctctcgcca aaacgccgcc 540
tctttccttc gtggcatcgc ccaccacgcc ctctcaacca gcccggcaca gagctccatt 600
ctggtcacca tggacagttg caaagagccg accaaggttc tacgcgcgta cacagctgac 660
ggcgtggttc cgttcgcgtt gtcggctctg aaatatgcga attccctgct cagaccagaa 720
gatgcagaga tagaaggggg tgaggttttc aaggtggatg actggtattt cctcagcgag 780
tggaactacg tacttgggag acacgaggca tccttgattc cacagtcgaa agatattaaa 840
ctgggggcac ctctggagag gatagtcgtg agaagagagg aaaagattcg ctttgggtgt 900
agctacaaat ataaccgagc agaaagagaa gagctcttcc gatctgctgg tgtaattgaa 960
gatgcacttt ggtcggatga aggctgtgat gtggcattct accagttgaa aatgattccg 1020
aattga 1026
<210> 135
<211> 873
<212> DNA
<213> Paecilomyces divaricatus
<400> 135
atgactatcc ttgtgctcgg tggccgcggc aagactgccc agcgtctcgc ctcgcttctc 60
gatgcggcca acattccctt cctgctgggg tcgagctcaa ccgctcatgt cagcccattc 120
aggctatcgt acttcaactg gttcgaagaa gaaacctatg gaaacccatt tgttcaagca 180
tccacggaag gcttggatgc tgtctcaacc gtgtatctag ttggaccccc ggtcatggac 240
atggttccgc caatgctcaa attcattgac ctcgcgcggt ccaggggtgt tgaaaagttc 300
gtgcttctca gcgccagtac agttgagaaa ggaggccact caatgggaga ggtccatgga 360
tacctagatt ctctgaaagg agtcaagtat gtcgcgttaa gaccgacttg gtttatggag 420
aacctcctgg aagaacccca tctttcgtgg atcaaacgac agaatacgat atattccgcg 480
acgggggacg gcaagattcc gtttatatcg gcagacgata ttgctcgggt agcatttcgg 540
gccttgacgg actcgaacac ccaagctaca gaatatctca tattagggcc agaattacta 600
tcctatggtc aggtggcgga catcctcacc tccgtccttg gcaggaaaat cactcatgtc 660
agtctctccg agccagattt tactcggctc ctagtggatg aggttggatt gcctcctgat 720
tttgcagcca tgctggccgg gatggaagta gatgtcaaga acggcggcga gaacagactc 780
agtaacagcg tcaagcaggt gacgggtata tcacctcaga ccttccatga ttttgcagtg 840
aaagagaagc agcactggac atatgatggc taa 873
<210> 136
<211> 870
<212> DNA
<213> Paecilomyces divaricatus
<400> 136
atgactgtta ccaagacctc caaaccagtg ctgcgccgcc tccccatcag tgctggcgcc 60
gatgccatct tccaagtcct ccaagaggac ggcgtgttga tcatcgaggg gttcctcaca 120
ccagaacagg tgaaaaagtt caacgcggaa gtcgaccctc atctcaagaa atgggagctc 180
ggccaaggtt cgatgcagga aggctatcta gccaacatga agcaactgag cagtctccca 240
ctgtttagca aaaccttccg ggacgatctt atgaatcatg acctcttgca cggcatctgc 300
aagaaggcct ttggtcctga ctccggagac tactggttga ccacgtcgtc ggtcttggag 360
acagatcctg gctaccccgg tcaagatctg caccgcgagc acgatgggat tccaatctgc 420
accaccctgg ggcgagacag tcctgaagcg atgctgaact tcctaacggc tctcacggac 480
ttcacggagg agaatggggc tacccgcgtt ctcccaggca gtcatctctg ggaggactac 540
agcaaatctg tctctcccga tcaggcgata ccggcagaga tgaaccctgg cgatgccgtg 600
ctgtttagtg ggaagactct gcatggcgct gggaaaaatc agagtaagga ttttctccgc 660
cgtggtttcc ccctcatcat gcaatcgtgc cagttcacgc cggttgaggc atccgtcgcc 720
attccaagat cccttgtgga gacgatgaca cctcttgcgc agaagatggt cggctggcga 780
tcggtatctg ccaagggtgt cgagatctgg acctatgact tgaaggatct tgcaaaggga 840
ttgaaattga aagctatagc tgctgagtga 870
<210> 137
<211> 1605
<212> DNA
<213> Aspergillus japonicus
<400> 137
atgactgctg ggcaaggcat caagacgggc aatgcctctg actgtgaagt ttaccgcacg 60
ctgagcgtcg ccctcgactt tgccaaccag gatgaggagc tctggtggca cagcaccgcg 120
cccatgtttg ctcagatgct gcagtccacc aactacaacc tccatgctca gtacaagcac 180
ctgctcatct acaagaaaaa cgtcatccct ttcctcgggg tttatcccac aaatgacaag 240
ccccggtggc taagcatcct gacccgatac ggcaccccct ttgagctgag cctgaactgc 300
tccggcccac tggtgcgcta cacctacgag cccatcaacg cggccacggg gacggcgcgc 360
gacccgttca acacccacgc tgtctgggac agtctggaac agctgatggc gctgcagagc 420
gggattgacc tggacctctt ccgccacttc aagaacgacc tcaccctcag cgctgaggaa 480
tctgagtacc tgtacaagaa caacctggtg ggcgagcaga tccggactca gaacaaactg 540
gcgctggatc tccaggacgg cgagttcgtg gtcaagacct acatctaccc ggccctgaag 600
tcgctcgcca cgggcaggtc catccacgag ctggtcttcg ggtctgcctt ccgctggtcc 660
aagcagtacc cggagctccg caagcccctg gacacactgg agcagtacgt ctactcgcgg 720
ggccccagca gcacggccag cccgcgtctc ctctcctgcg acctgatcga ccccaccaag 780
tcccgcatca agatctacct gctggagcgg atggtcaccc tggaggccct ggaggacctc 840
tggaccatgg ggggcgagcg caccgacgcc tcgaccctgg cggggctcga gatgatccgc 900
gagctgtggg agctgatccg cctgcccgcg ggcttgcagt cgtatccggc cccgtatctg 960
cccatcggga ccatcccgga cgagcagctg cccctgatgg ccaactacac catccaccac 1020
gacgaccccg ttccggagcc gcaggtctac ttcaccacct tcggccgcaa cgacatgcag 1080
atcgccgacg cgctggccac cttcttcgag cgccgcggct ggcacgagat ggcgcgccag 1140
tacaaggccg agctgtgcag ccactacccg cacgccgacc acgagaccct gaactacctg 1200
cacgcctaca tctccttctc ctaccgcaag aacaaaccct acctcagcgt gtatctgcag 1260
tcgctggaga cgggcgactg ggtcacttgt aagctctccc cttccgtgtt cctttcctgc 1320
acacttcccc accgtggata catccctgat gatctgacct gttcgcgcag catccttcaa 1380
ctctgtccat gtggaccccg gcctctcagc caccgtccaa gagctgtcca agctgaccaa 1440
gaccgccggg accaccgttc gcgagacgaa actcccactc accccggatg gatcggagcc 1500
cggcgtcatc acccagtact aggaaatcat ctcgttggac cggatttgcc agcttcccgc 1560
cggggagttg caagtgcttt gtatgtaggt aggggaacta gatga 1605
<210> 138
<211> 1239
<212> DNA
<213> Aspergillus japonicus
<400> 138
atgacgatcg acccctcctc ccccggggcg cccgcctcga agctcttcca acccatgaag 60
ataggcctat gtcacctcca gcaccgcctc atcatggccc ccaccacccg ctaccgcgcc 120
gacagcaccg cgacgcccct ccccttcgtg caagaatact acgcccaacg cgccgccgtt 180
ccggggacac tcctgatcac cgaagccacc gacatctccc cgcaggcggc cggagaaccg 240
cacatcccag gcctgtacag cgcaacccag atcgccgcgt ggcgggggat cgtctccgcc 300
gtgcaccgcc ggggctgcta catcttctgc cagctgtggg cgaccggacg ggcggcggac 360
cccgccctgt tgaaggagaa ggggttccct ctcgtctcga gcagcgatgt gcccatcgag 420
actgaatcgc agcagccgca gccgctgacg caggccgaga tcgaggggta catcgcggac 480
ttcgtgacgg cggcaagggt agcggtccac gaggtggggt tcgatggggt ggagctgcac 540
ggcgccaacg gatacctcat cgatcagttc acgcagtggc cgtgcaacca gcggacggat 600
tgctggggcg ggagcatcga gaaccgcgcg cggtttgcgc tggcggtcac tcgcgcggtg 660
gtggatgccg tcggggcgga gcgcgtcggg gttaagctgt cgccctggag tcagtacgcc 720
gggatggggg tgatggctga tttggtgccg cagtttgagt tcttgatcaa aaggttacgg 780
gaggagtaca atgatggtcc tgggctggcg tacttgcatc tggcgaattc aaggtggttg 840
gatgaggaga cttcccatcc ggatccgcat catgaggtct tcgtgcgcgc gtggaggggg 900
tcgggcaggg atggtattga ggcgaagaaa gaggcgcctc cggtgatcct ggccggcggc 960
tatgatgctg tctcggcgcc cgaggtcgct gataaggtgt tcaaggaaga ggacaatgtc 1020
gccgtcgcgt ttgggcgata ttttatctcg acgcccgacc tgccgttccg catgcaacgg 1080
ggcattccat tgcagaagta tgatcgcgcc tcgttctata cctcgctttt gaaagaaggt 1140
tatctggatt atccgtacag tgcggagtat gttgcggagt ttgggcagag gctgaagaag 1200
aaggaggcgg cggcgacagg gtcgatatgt cagggttga 1239
<210> 139
<211> 1530
<212> DNA
<213> Aspergillus japonicus
<400> 139
atggcgcccg atagcaagcc gacctacacc acggcaaatg gctgtccctt ccacaagcct 60
gagggtcctc cggcggatgg aaggacgctg gcactcagcg accaccatct ggtcgatacc 120
ctcgcccact tcaaccggga gaagatcccc gagcgagtgg tgcatgctaa aggagcggga 180
gcctatggcg aatttgaggt gacggccgac atctcggata tctgcgatat cgacatgctg 240
gtgggagtgg gcaagaagac cccctgtgtg acgcggttct cgacgactgg cctcgaacgc 300
gggtccaacg agggaatgcg cgacctgaaa ggcatggcct gcaagttcta caccaccgag 360
ggcaactggg actgggtgat gctcaacttc cccttcttct tcatccgcga ccccgtcaag 420
ttccccagcc tgatgcacgc ccagcgccgg gatccgcaga ccaacctgct gaaccccaac 480
atgtactggg actgggtgac caacaaccac gagtcgctgc acatggtcct gctgcagttc 540
agcgacttcg ggaccatgtt caactggcgg tccatgagcg gctacatggc ccacgcgtac 600
aaatgggtga agcccgacgg gtcgttcaaa tacgtccaca tcttcctctc ctccgatcgc 660
gggcccaact tcaccgacgg ccagcaggcc aagggcacga acgacctgga tccggaccac 720
gccacccgcg acctgtacga ggccatcgag cgcggcgagt atccgacgtg gacggcgagc 780
gtgcaggtgg tcgaccccaa ggacgcgccg aacctggggt acaacatcct ggacgtgacc 840
aagcactgga acctgggcac gtaccccaag gacgtgacgc tgatcccgcc caaggtcttc 900
ggcaagctga cgctcaagcg caacccggcc aactacttcg ccgagatcga gcagctggcc 960
tactccccct cgaacatggt gcccggggtg gccccctcgg aggacccgat cctccaggcg 1020
cgcatcttcg cctacccgga cgcccagcgg taccggctgg gcgccaacca ccagcagatc 1080
cccgtcaacc ggtccgccca caccttcaac cccatcgcgc gcgacggcca gggcaccttc 1140
gacgccaact acggcgccca cccgggcttt ctgacccagc agcagcccgt ccgcttcgcc 1200
gagccccgcg agcccgaccc caagtacaac gagtggctca aggagatcca gtccaagtcc 1260
tggctccaga ccaccgagca cgactacaag ttcgcccgcg acttctacga ggtcctgccg 1320
gacttccgcg ggcaggagtt ccaggacacc atggtccaga atatggtcga gtccgtcgcg 1380
cagacgcgcg cggagatcca aaagcaggtc tacgagacct ggaagctggt gagtccagcg 1440
ctggcggcgc gaatccagaa aggggtggag acgctgctgc agaagagtga ggcgccggcg 1500
gaggagtctt tcatcccggc aagattgtag 1530
<210> 140
<211> 789
<212> DNA
<213> Aspergillus japonicus
<400> 140
atgaccgtcg tcaactcgcg catctttgca gtcaccggcg gcgcctccgg cattggcgcg 60
gccacctgcc gcctcctcgc cgcgcgcggc gcagcggtca tctgcatcgg ggacatctcg 120
cccaagggct tcgagagcgt caccagcgac atccaggccc tcaacccggc caccaaggtg 180
cactgcaccc ggctggacgt ctcctcgtcc gccgaggttg acgcctggct acagaccatc 240
gtgcagacct tcggggactt gcacgggctg gccaacgtcg cgggcatcgc ccagccggcg 300
ggcgcccgcc aggccccggc cctgctcgag gagacggacg aggaatggca gcgcgtgttc 360
aaggtcaacc tggacggggt cttctactgc acgcgcgcag gtatccgcgc gatgaaggcc 420
cttccggggc ccaaggaccg cgccatcgtc aacatcggta gcatcgccgc gtttattcac 480
ttgccggatg cctacgccta cgggacctcc aagggcgcct gcgcgtactt caccagctgt 540
gcggccatgg atgcctggcc cctggggatt cggatcaaca gtgtctcgcc gggtatcacc 600
gacaccccgg ctctgccgca gttcatgccc caggcgaagt ccatcgatga ggtcaaggaa 660
gcgtacatca aggaggggtt ccctatcatt cagcctgagg atgtggccag gacgattgtc 720
tggctgctca gcgaggattc ccgcccggtg tatggggcca atatcaatgt tggggctgct 780
actccttga 789
<210> 141
<211> 1866
<212> DNA
<213> Aspergillus japonicus
<400> 141
atgggtcagt cacgagggat cttgggaggc gtgaggcaac ttatcctggt tattctggtt 60
ggggcatacc tcagccggct ttcagcggtg gatgacgacc gccacgactg tcgatgtcgg 120
ccgggggagc catgctggcc cacggtcagc cactggagta ctctgaacga gtccatcggc 180
ggtgctctgg ctcatgtcaa acccatcgct cacgtctgtc accagtctgg tcaggactcg 240
tcggcttgcg agcaggtctt gcaagagagt cttgacagca agtggcgggc gtcgcacacg 300
ggcgcattgc aagactgggt ctgggaaggt ggcgcggaat ccaaccagac atgttactac 360
ctgcggtccg gtccggctgg atcatgccac cagggccgga ttcccctcta ctccgcggct 420
gtcaagtcgg ccagcgacgt gcagaaggtc gtggacttca ctcgtcaaca caatttacga 480
ttagtgatcc gcaacaccgg ccacgatggg agcggccgat ccagtggacc ggactctgtc 540
gagatccaca cccaccatct gaacagcgtg caatatcacc ccaacttccg ccccgctgga 600
tcttctgagc gccagtcggc tccaggacag ccagcggtga cagtaggcgc cggaatcctg 660
cttggggacc tctacgcccg cggcgcctcg gagggatgga tcgtagtggg cggggagtgt 720
cccaccgtcg gggcggccgg cggattcctc cagggcggag gagtatcgag ttggctgagc 780
tacgcgcatg gattggcagt ggataatgtg cttgagtatg aagttgtgac ggccaaggga 840
gagattgtca tcgccaacgc ccatcaaaac cccgatctct tctgggcact gcgcggggga 900
ggaggaggca cctttggcgt cgtgacccag gccacgcttc aagtccaccc cgacctcccc 960
gtcagcgtcg ccgacgccgt ggtgacgggc tcccgcgccg acgcgacctt ctggtcccac 1020
ggcgtcgccg ccctcctgcg agcgctacaa ttcctcaaca accacggcac ggcaggccag 1080
ttcatcctcc gcaacaccga cgacgacacg gtgcaagcca gtctgacgat gtacttctcg 1140
aacctgaccg tccccgccgt cgccgacgag cgcatggacc cgctccgccg cgccctcgaa 1200
cacaacggac acccctacca gctcacctcg cggttcctgc cccagatcag cagcaccttc 1260
cggcacaccg cggatcgcta cccggaggac tacggaatcc tgatgggctc ggtcctggtg 1320
tcgctggacc tgttcaattc cgcgacgggc cccgcggcgc tggcgcagca cttcgcgcgg 1380
ctcccgatga cccccgacga tctcctgttc accagcaacc tgggcgggcg agtgtcctcc 1440
ctcaaccgcg acccggccag cacagccatg catcccggct ggcgtgacgc cgcgcagttg 1500
ctcaacttcg tccgcggggt cggggcccct tctctcgcgg ctaaggccac cgccctccac 1560
gagctgcaga cggtccagat ggcgaggctg tatgagattg aaccggcctt ccagatctcg 1620
taccgcaacc tgggagatcc ctcggagcgc cgcagtcggg aggtctattg ggggtcgaac 1680
tacgcgcggt tagtggaggt caagcggagg tgggatccgg agggactgtt cttcagcaag 1740
ctggggattg atggtgatgc atgggacgcg gaaggaatgt gtcgtcagac gcgccaaggg 1800
gcgtggaatg tggcggtcaa atgggtccag agcttccttg ggtctttgtc tgcttctgtg 1860
gtgtag 1866
<210> 142
<211> 1020
<212> DNA
<213> Aspergillus japonicus
<400> 142
atgggcagca tcaatccccc gattttggac atccgccagt cgaccttcga gacctccatc 60
ccccaacaag taaccgaggg actcaccaaa gaccccaaaa ccctgccggc ccttcttttc 120
tacagcggcg atggcatcca gcactggacg cgccactcgt gcgccccgga tttctacccc 180
cgccgcgagg agatcaacat cctccacagc gaagcggctc acatggcggc ctcgataagc 240
caggacagtg ttgtggtgga tctgggcagc gcatccctcg ataaagtcct ccccttcctg 300
cacgccctcg aggcccaaca gaagcgcgtt cactactacg ccctcgacct gagctacccg 360
gtgctcgcct cgacgctgtc cgagatcccc gtggcgcagt tccagtatgt ccagatcgcc 420
gcgctgcacg gcaccttcga ggacgggctg cagtggctgc acgactcgcc cgaggtccgc 480
gagcgccccc accacctgct gctgttcggc ctcacgatcg gcaacttctc acggcccaac 540
gccgccgcct ttttacggaa tatcgccgac cgggcgctgg cggcgtcccc cgcggagagc 600
tccatcctcc tcaccctgga cagctgcaag atgccgacca aggtgctgcg cgcgtacacg 660
gcggaagggg tggtgccttt cgcgctgacc tcgttgagct atgccaatca gctcttctgg 720
ccgggtgggg agggaagggt gtttgatgaa gaggaatggt atttccacag tgagtggaac 780
ttcgcgctgg ggcgccacga ggcggcgctc atcccccggg atcgcgatat caccctcggg 840
ccgccgttgg agcaggtcgt cgtgcgccgg ggtgagaagg tgaggtttgg ctgcagctac 900
aagtatagtg ctgcggagcg ggagcatttg ttcgctgggg cggggttgga gaatgtggct 960
tcctggtcgg ccaagggctg tgatgttgct ttttatcagt tgaagatgcg gcccgagtga 1020
<210> 143
<211> 855
<212> DNA
<213> Aspergillus japonicus
<400> 143
atgaccatcc tcgtcctcgg cggccgcggc aagaccgcca gccgcctctc agccctcctg 60
cacgaagccc agatcccctt cgtcgtgggc acctcctcca cggccccgat cgccacctac 120
cgaaccgccc acttcgactg gctgaacaaa gacacctgga ccaacgcgtg ggaccaggcc 180
ccggagccca tcaccgctat ctacctcgta ggcgcccacg ccccggagtt cgtgcacccg 240
atgttcgagt tcatcgacct ggcgcacagc cggggcgtgc ggcggtacgc gctcgtcagc 300
gcgagcaaca ccgaaaaggg ggatccgatg atgggggagg cgcacgcgca cctggactcg 360
aaagaggggg tcaagtatgt ggtgttgcgg ccgacgtggt ttatggaaaa ccacatcgaa 420
gacccccacc tcagctggat caagacggag aacaagatct actcggccac gggcccgggc 480
aagatcccct tcgtctcggc ggatgatatc gcccgcgtcg ccttccacgc cctgacgcag 540
ccgctcaccc agaaggagca gcagtacatg gttctgggcc cggatctgct gtcctatgct 600
gatatggccg agatcctcac cgacctcctg ggccggaaga ttacgcacgt caccctgccc 660
gccgagaagt tcggccagtt cctggtcgac acggccgggt tgcccccgga ctttgcggag 720
atgctggcga cgatggaggc ggcggtggcg acgggggcgg aggagcgggt cagccaagat 780
gtcgagaagg tgaccgggcg gccgccgcgg cggtttgtgg attacgcgag ggagaatcgc 840
gagcattggt tgtga 855
<210> 144
<211> 882
<212> DNA
<213> Aspergillus japonicus
<400> 144
atgaccatca ccgagacctc caagccgacg ctgcgccgca tcccccggag cgccggcgac 60
gaggccatct tccaagtcct gcaggaagac ggagtcgtcg tgatcgaagg attcatgagc 120
gcggaccagg tccgccgctt caacggggag atcgacccgc acatgaagca gtgggaactg 180
ggccagaagt cgtaccagga gagctacctg gcggggatgc gacagctgag cagcctgccg 240
ctgttcagca agctcttccg ggacgagctg atgaacgacg agctcctgca cgggctgtgc 300
aagcggctgt tcgggcccga gtccggcgac tactggctga cgacctcctc ggtgctggag 360
acagagccgg gctaccacgg gcaggagctg caccgcgagc acgacgggat ccccatctgc 420
accaccctgg gccgccagag ccccgagtcc atgctcaact tcctcaccgc gctgaccgac 480
ttcaccgcgg agaacggcgc cacccgcgtc ctgccgggca gtcacctctg ggaggacttc 540
agcgccccgc cccccaaggc cgacaccgcc atccccgccg tcatgaaccc cggcgatgcc 600
gttctcttca ccggcaagac cctgcacggc gccggcaaga acaacaccac cgacttcctg 660
cgccgcggct tccccctgat catgcagtcg tgccagttca cccctgtcga ggcctcggtg 720
gccctgcccc gtgagctggt cgagaccatg acccccctgg cccagaagat ggtcggctgg 780
cggaccgtct ccgctaaggg ggtggatatc tggacgtatg atctgaagga tctggcgaca 840
ggaattgatc tcaagtcgaa ccaggtggcg aaaaaggcgt ga 882
<210> 145
<211> 31
<212> PRT
<213> Artificial Sequence
<220>
<223> DmaW pattern 1_SeqID 145
<220>
<221> MISC_FEATURE
<222> (2)..(2)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (12)..(12)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (13)..(13)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (14)..(14)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (15)..(15)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (17)..(17)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (18)..(18)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (20)..(20)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (22)..(22)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (25)..(25)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (28)..(28)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (29)..(29)
<223> Xaa can be any naturally occurring amino acid
<400> 145
Ile Xaa Thr Gln Asn Lys Leu Ala Leu Asp Leu Xaa Xaa Xaa Xaa Phe
1 5 10 15
Xaa Xaa Lys Xaa Tyr Xaa Tyr Pro Xaa Leu Lys Xaa Xaa Thr Gly
20 25 30
<210> 146
<211> 91
<212> PRT
<213> Artificial Sequence
<220>
<223> DmaW pattern 2
<220>
<221> MISC_FEATURE
<222> (3)..(3)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (8)..(9)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (11)..(11)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (14)..(14)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (18)..(19)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (21)..(22)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (24)..(24)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (26)..(28)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (34)..(34)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (37)..(39)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (41)..(42)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (44)..(45)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (48)..(49)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (55)..(55)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (57)..(65)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (68)..(70)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (72)..(73)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (75)..(76)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (77)..(77)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (78)..(78)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (80)..(80)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (83)..(83)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (85)..(85)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (87)..(87)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (89)..(90)
<223> Xaa can be any naturally occurring amino acid
<400> 146
Arg Leu Xaa Ser Cys Asp Leu Xaa Xaa Pro Xaa Ser Arg Xaa Lys Ile
1 5 10 15
Tyr Xaa Xaa Glu Xaa Xaa Val Xaa Leu Xaa Xaa Xaa Glu Asp Leu Trp
20 25 30
Thr Xaa Gly Gly Xaa Xaa Xaa Asp Xaa Xaa Thr Xaa Xaa Gly Leu Xaa
35 40 45
Xaa Leu Arg Glu Leu Trp Xaa Leu Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
50 55 60
Xaa Tyr Pro Xaa Xaa Xaa Leu Xaa Xaa Gly Xaa Xaa Pro Xaa Glu Xaa
65 70 75 80
Leu Pro Xaa Met Xaa Asn Xaa Thr Xaa Xaa His
85 90
<210> 147
<211> 444
<212> PRT
<213> Artificial Sequence
<220>
<223> DmaW consensus
<220>
<221> misc_feature
<222> (2)..(4)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (6)..(10)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (13)..(13)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (17)..(18)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (22)..(25)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (27)..(27)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (39)..(39)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (45)..(45)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (47)..(48)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (50)..(50)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (53)..(53)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (61)..(62)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (72)..(75)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (96)..(96)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (98)..(98)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (113)..(113)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (120)..(120)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (124)..(124)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (127)..(128)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (130)..(132)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (134)..(134)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (142)..(142)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (146)..(147)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (151)..(153)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (159)..(160)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (163)..(163)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (166)..(167)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (182)..(183)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (197)..(197)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (203)..(203)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (207)..(207)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (212)..(212)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (216)..(218)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (222)..(223)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (226)..(227)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (230)..(230)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (233)..(233)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (237)..(246)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (249)..(249)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (259)..(259)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (262)..(262)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (278)..(278)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (291)..(291)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (293)..(293)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (297)..(297)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (307)..(307)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (309)..(310)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (313)..(313)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (316)..(317)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (320)..(320)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (324)..(324)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (327)..(327)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (330)..(330)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (344)..(344)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (347)..(347)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (356)..(356)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (362)..(366)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (378)..(379)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (382)..(383)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (386)..(387)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (389)..(391)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (395)..(395)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (397)..(397)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (399)..(399)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (413)..(413)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (432)..(439)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (441)..(443)
<223> Xaa can be any naturally occurring amino acid
<400> 147
Met Xaa Xaa Xaa Asn Xaa Xaa Xaa Xaa Xaa Val Tyr Xaa Thr Leu Ser
1 5 10 15
Xaa Xaa Phe Asp Phe Xaa Xaa Xaa Xaa Gln Xaa Leu Trp Trp His Ser
20 25 30
Thr Ala Pro Met Phe Ala Xaa Met Leu Gln Thr Ala Xaa Tyr Xaa Xaa
35 40 45
His Xaa Gln Tyr Xaa His Leu Gly Ile Tyr Lys Lys Xaa Xaa Ile Pro
50 55 60
Phe Leu Gly Val Tyr Pro Thr Xaa Xaa Xaa Xaa Arg Trp Leu Ser Ile
65 70 75 80
Leu Thr Arg Tyr Gly Thr Pro Phe Glu Leu Ser Leu Asn Cys Ser Xaa
85 90 95
Ser Xaa Val Arg Tyr Thr Tyr Glu Pro Ile Asn Ala Ala Thr Gly Thr
100 105 110
Xaa Lys Asp Pro Phe Asn Thr Xaa Ala Ile Trp Xaa Ser Leu Xaa Xaa
115 120 125
Leu Xaa Xaa Xaa Gln Xaa Gly Ile Asp Leu Glu Trp Phe Xaa Tyr Phe
130 135 140
Lys Xaa Xaa Leu Thr Leu Xaa Xaa Xaa Glu Ser Ala Tyr Leu Xaa Xaa
145 150 155 160
Asn Asn Xaa Leu Val Xaa Xaa Gln Ile Lys Thr Gln Asn Lys Leu Ala
165 170 175
Leu Asp Leu Lys Gly Xaa Xaa Phe Val Leu Lys Thr Tyr Ile Tyr Pro
180 185 190
Ala Leu Lys Ser Xaa Ala Thr Gly Lys Ser Xaa His Glu Leu Xaa Phe
195 200 205
Gly Ser Val Xaa Arg Leu Ser Xaa Xaa Xaa Pro Ser Ile Xaa Xaa Pro
210 215 220
Leu Xaa Xaa Leu Glu Xaa Tyr Val Xaa Ser Arg Gly Xaa Xaa Xaa Xaa
225 230 235 240
Xaa Xaa Xaa Xaa Xaa Xaa Ser Thr Xaa Ser Pro Arg Leu Leu Ser Cys
245 250 255
Asp Leu Xaa Asp Pro Xaa Lys Ser Arg Val Lys Ile Tyr Leu Leu Glu
260 265 270
Arg Met Val Ser Leu Xaa Ala Met Glu Asp Leu Trp Thr Leu Gly Gly
275 280 285
Arg Arg Xaa Asp Xaa Ser Thr Leu Xaa Gly Leu Glu Met Ile Arg Glu
290 295 300
Leu Trp Xaa Leu Xaa Xaa Leu Pro Xaa Gly Leu Xaa Xaa Tyr Pro Xaa
305 310 315 320
Pro Tyr Leu Xaa Leu Gly Xaa Ile Pro Xaa Glu Gln Leu Pro Leu Met
325 330 335
Ala Asn Tyr Thr Leu His His Xaa Asp Pro Xaa Pro Glu Pro Gln Val
340 345 350
Tyr Phe Thr Xaa Phe Gly Met Asn Asp Xaa Xaa Xaa Xaa Xaa Ala Leu
355 360 365
Thr Thr Phe Phe Glu Arg Arg Gly Trp Xaa Xaa Met Ala Xaa Xaa Tyr
370 375 380
Lys Xaa Xaa Leu Xaa Xaa Xaa Tyr Pro His Xaa Asp Xaa Glu Xaa Leu
385 390 395 400
Asn Tyr Leu His Ala Tyr Ile Ser Phe Ser Tyr Arg Xaa Asn Lys Pro
405 410 415
Tyr Leu Ser Val Tyr Leu His Ser Phe Glu Thr Gly Asp Trp Pro Xaa
420 425 430
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Phe Xaa Xaa Xaa Arg
435 440
<210> 148
<211> 64
<212> PRT
<213> Artificial Sequence
<220>
<223> EasA patern 1
<220>
<221> misc_feature
<222> (2)..(2)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (5)..(8)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (13)..(15)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (17)..(18)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (20)..(24)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (26)..(29)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (31)..(40)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (42)..(47)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (50)..(54)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (56)..(57)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (60)..(62)
<223> Xaa can be any naturally occurring amino acid
<400> 148
Tyr Xaa Gln Arg Xaa Xaa Xaa Xaa Gly Thr Leu Leu Xaa Xaa Xaa Ala
1 5 10 15
Xaa Xaa Ile Xaa Xaa Xaa Xaa Xaa Gly Xaa Xaa Xaa Xaa Pro Xaa Xaa
20 25 30
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Trp Xaa Xaa Xaa Xaa Xaa Xaa Val
35 40 45
His Xaa Xaa Xaa Xaa Xaa Ile Xaa Xaa Gln Leu Xaa Xaa Xaa Gly Arg
50 55 60
<210> 149
<211> 45
<212> PRT
<213> Artificial Sequence
<220>
<223> EasA patern 2
<220>
<221> misc_feature
<222> (2)..(5)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (9)..(9)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (11)..(11)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (13)..(14)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (18)..(18)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (20)..(20)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (24)..(24)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (26)..(28)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (30)..(30)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (32)..(32)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (34)..(35)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (38)..(41)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (43)..(43)
<223> Xaa can be any naturally occurring amino acid
<400> 149
Ala Xaa Xaa Xaa Xaa Phe Asp Gly Xaa Glu Xaa His Xaa Xaa Ala Asn
1 5 10 15
Gly Xaa Leu Xaa Asp Gln Phe Xaa Gln Xaa Xaa Xaa Asn Xaa Arg Xaa
20 25 30
Asp Xaa Xaa Gly Gly Xaa Xaa Xaa Xaa Arg Xaa Arg Phe
35 40 45
<210> 150
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> EasA patern 3
<220>
<221> misc_feature
<222> (3)..(5)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (7)..(7)
<223> Xaa can be any naturally occurring amino acid
<400> 150
Gly Arg Xaa Xaa Xaa Ser Xaa Pro Asp Leu Pro Phe
1 5 10
<210> 151
<211> 496
<212> PRT
<213> Artificial Sequence
<220>
<223> EasC consensus
<220>
<221> misc_feature
<222> (2)..(4)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (6)..(7)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (17)..(18)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (22)..(25)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (27)..(30)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (32)..(32)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (34)..(34)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (36)..(36)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (41)..(41)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (70)..(70)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (77)..(77)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (89)..(89)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (120)..(120)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (128)..(128)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (131)..(131)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (144)..(144)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (158)..(158)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (169)..(169)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (179)..(179)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (189)..(189)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (226)..(227)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (229)..(230)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (232)..(233)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (236)..(237)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (241)..(241)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (271)..(271)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (288)..(288)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (291)..(291)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (294)..(295)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (300)..(300)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (311)..(311)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (356)..(356)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (359)..(360)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (365)..(366)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (368)..(369)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (371)..(371)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (373)..(374)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (378)..(382)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (388)..(388)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (390)..(390)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (392)..(392)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (394)..(397)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (399)..(399)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (402)..(403)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (406)..(408)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (412)..(413)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (417)..(417)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (419)..(419)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (421)..(421)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (424)..(427)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (429)..(429)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (439)..(440)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (443)..(444)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (446)..(446)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (452)..(452)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (455)..(455)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (459)..(459)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (464)..(466)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (474)..(474)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (477)..(477)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (481)..(481)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (485)..(485)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (489)..(489)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (493)..(493)
<223> Xaa can be any naturally occurring amino acid
<400> 151
Ala Xaa Xaa Xaa Ser Xaa Xaa Tyr Thr Thr Ser Asn Gly Cys Pro Val
1 5 10 15
Xaa Xaa Pro Glu Gly Xaa Xaa Xaa Xaa Gly Xaa Xaa Xaa Xaa Leu Xaa
20 25 30
Leu Xaa Asp Xaa His Leu Ile Asp Xaa Leu Ala His Phe Asn Arg Glu
35 40 45
Lys Ile Pro Glu Arg Ala Val His Ala Lys Gly Ala Gly Ala Tyr Gly
50 55 60
Glu Phe Glu Val Thr Xaa Asp Ile Ser Asp Ile Cys Xaa Ile Asp Met
65 70 75 80
Leu Leu Gly Val Gly Lys Lys Thr Xaa Cys Val Thr Arg Phe Ser Thr
85 90 95
Thr Gly Leu Glu Arg Gly Ser Ala Asp Ser Val Arg Asp Leu Lys Gly
100 105 110
Met Ala Val Lys Phe Tyr Thr Xaa Glu Gly Asn Trp Asp Trp Val Xaa
115 120 125
Leu Asn Xaa Pro Met Phe Phe Ile Arg Asp Pro Ser Lys Phe Pro Xaa
130 135 140
Leu Ile His Ala Gln Arg Arg Asp Pro Gln Thr Asn Leu Xaa Asn Pro
145 150 155 160
Ser Met Phe Trp Asp Phe Val Thr Xaa Asn His Glu Ala Leu His Met
165 170 175
Val Met Xaa Gln Phe Ser Asp Phe Gly Thr Met Phe Xaa Tyr Arg Ser
180 185 190
Met Ser Gly Tyr Val Gly His Ala Tyr Lys Trp Val Met Pro Asp Gly
195 200 205
Ser Phe Lys Tyr Val His Ile Phe Leu Ser Ser Asp Gln Gly Pro Asn
210 215 220
Phe Xaa Xaa Gly Xaa Xaa Ala Xaa Xaa Ile Ala Xaa Xaa Asp Pro Asp
225 230 235 240
Xaa Ala Thr Arg Asp Leu Tyr Glu Ala Ile Glu Arg Gly Glu Tyr Pro
245 250 255
Ser Trp Thr Ala Asn Val Gln Val Val Asp Pro Glu Asp Ala Xaa Lys
260 265 270
Leu Gly Phe Asn Ile Leu Asp Val Thr Lys His Trp Asn Leu Gly Xaa
275 280 285
Tyr Pro Xaa Asp Leu Xaa Xaa Ile Pro Ser Arg Xaa Phe Gly Lys Leu
290 295 300
Thr Leu Asn Lys Asn Pro Xaa Asn Tyr Phe Ala Glu Ile Glu Gln Leu
305 310 315 320
Ala Phe Ser Pro Ser His Leu Val Pro Gly Val Glu Pro Ser Glu Asp
325 330 335
Pro Ile Leu Gln Ala Arg Leu Phe Ala Tyr Pro Asp Ala Gln Arg Tyr
340 345 350
Arg Leu Gly Xaa Asn His Xaa Xaa Ile Pro Val Asn Xaa Xaa Lys Xaa
355 360 365
Xaa Phe Xaa Pro Xaa Xaa Arg Asp Gly Xaa Xaa Xaa Xaa Xaa Gly Asn
370 375 380
Tyr Gly Ala Xaa Pro Xaa Tyr Xaa Ser Xaa Xaa Xaa Xaa Met Xaa Phe
385 390 395 400
Ala Xaa Xaa Lys Ser Xaa Xaa Xaa Pro Glu His Xaa Xaa Trp Leu Ser
405 410 415
Xaa Val Xaa Ser Xaa Ser Trp Xaa Xaa Xaa Xaa Glu Xaa Asp Tyr Lys
420 425 430
Phe Ala Arg Glu Phe Trp Xaa Xaa Leu Pro Xaa Xaa Arg Xaa Gln Glu
435 440 445
Phe Gln Asp Xaa Met Val Xaa Asn Met Ala Xaa Ser Val Ala Gln Xaa
450 455 460
Xaa Xaa Glu Leu Arg Lys Lys Val Tyr Xaa Thr Phe Xaa Leu Val Ala
465 470 475 480
Xaa Asp Leu Ala Xaa Arg Val Lys Xaa Gly Thr Glu Xaa Leu Val Ala
485 490 495
<210> 152
<211> 266
<212> PRT
<213> Artificial Sequence
<220>
<223> EasD consensus
<220>
<221> misc_feature
<222> (28)..(28)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (42)..(42)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (45)..(45)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (48)..(48)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (75)..(75)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (78)..(78)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (112)..(112)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (114)..(114)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (117)..(117)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (121)..(121)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (143)..(145)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (147)..(147)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (182)..(182)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (191)..(191)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (197)..(197)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (202)..(202)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (213)..(213)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (215)..(215)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (223)..(223)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (225)..(225)
<223> Xaa can be any naturally occurring amino acid
<400> 152
Met Ala Ser Val Ser Ser Lys Ile Phe Ala Ile Thr Gly Gly Ala Ser
1 5 10 15
Gly Ile Gly Ala Ala Thr Cys Arg Leu Leu Ala Xaa Arg Gly Ala Ala
20 25 30
Val Ile Cys Val Gly Asp Val Ser Ser Xaa Asn Phe Xaa Ser Leu Xaa
35 40 45
Lys Ser Ile Lys Glu Ile Asn Pro Ser Thr Lys Val His Cys Thr Val
50 55 60
Leu Asp Val Ser Ser Ser Ser Glu Val Asp Xaa Trp Leu Xaa Asp Ile
65 70 75 80
Ile Ser Thr Phe Gly Asp Leu His Gly Ala Ala Asn Val Ala Gly Ile
85 90 95
Ala Gln Gly Ala Gly Leu Arg Gln Thr Pro Thr Ile Leu Glu Glu Xaa
100 105 110
Asp Xaa Glu Trp Xaa Arg Ile Phe Xaa Val Asn Leu Asp Gly Val Phe
115 120 125
Tyr Ser Thr Arg Ala Gln Val Arg Ala Met Lys Asp Leu Pro Xaa Xaa
130 135 140
Xaa Gly Xaa Asp Arg Ser Ile Val Asn Val Ala Ser Ile Ala Ala Phe
145 150 155 160
Ser His Met Pro Asp Val Tyr Ala Tyr Gly Thr Ser Lys Ala Ala Cys
165 170 175
Ala Tyr Phe Thr Thr Xaa Cys Val Ala Ala Asp Val Phe Pro Xaa Gly
180 185 190
Ile Arg Val Asn Xaa Val Ser Pro Gly Xaa Ile Thr Asn Thr Pro Leu
195 200 205
Leu Pro Gln Phe Xaa Pro Xaa Ala Lys Ser Leu Asp Glu Val Xaa Glu
210 215 220
Xaa Tyr Lys Lys Glu Gly Phe Ser Val Ile Glu Ala Asp Asp Val Ala
225 230 235 240
Arg Thr Ile Val Trp Leu Leu Ser Glu Asp Ser Arg Pro Val Tyr Gly
245 250 255
Ala Asn Ile Asn Val Gly Ala Cys Met Pro
260 265
<210> 153
<211> 609
<212> PRT
<213> Artificial Sequence
<220>
<223> EasE consensus
<220>
<221> misc_feature
<222> (2)..(3)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (6)..(12)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (14)..(16)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (18)..(26)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (34)..(34)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (39)..(41)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (43)..(43)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (45)..(45)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (47)..(47)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (52)..(52)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (54)..(55)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (61)..(61)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (65)..(65)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (67)..(67)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (70)..(70)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (74)..(75)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (78)..(78)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (92)..(92)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (102)..(102)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (113)..(113)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (115)..(116)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (118)..(120)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (138)..(138)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (142)..(142)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (163)..(163)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (184)..(184)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (187)..(187)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (192)..(192)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (195)..(198)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (203)..(203)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (220)..(220)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (224)..(224)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (255)..(256)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (272)..(272)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (322)..(322)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (324)..(325)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (327)..(327)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (333)..(333)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (337)..(337)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (357)..(360)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (369)..(369)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (378)..(378)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (380)..(380)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (383)..(383)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (388)..(388)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (391)..(391)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (394)..(395)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (398)..(398)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (401)..(401)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (405)..(405)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (408)..(409)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (412)..(412)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (415)..(416)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (424)..(424)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (429)..(429)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (437)..(437)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (440)..(440)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (446)..(446)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (450)..(451)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (454)..(454)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (460)..(460)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (477)..(478)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (503)..(503)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (512)..(512)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (516)..(516)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (520)..(520)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (531)..(531)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (554)..(554)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (556)..(556)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (562)..(562)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (583)..(583)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (593)..(596)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (598)..(599)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (601)..(601)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (603)..(603)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (605)..(606)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (608)..(608)
<223> Xaa can be any naturally occurring amino acid
<400> 153
Met Xaa Xaa Leu Val Xaa Xaa Xaa Xaa Xaa Xaa Xaa Leu Xaa Xaa Xaa
1 5 10 15
Leu Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Cys Arg Cys Arg Pro Trp
20 25 30
Glu Xaa Cys Trp Pro Ser Xaa Xaa Xaa Trp Xaa Ser Xaa Asn Xaa Ser
35 40 45
Ile Asp Gly Xaa Leu Xaa Xaa Leu Arg Pro Ile Ala Xaa Val Cys His
50 55 60
Xaa Pro Xaa Phe Asp Xaa Ser Ala Cys Xaa Xaa Leu Leu Xaa Leu Ser
65 70 75 80
Arg Asp Ser Gly Trp Arg Ala Ser Asn Pro Gly Xaa Leu Gln Asp Trp
85 90 95
Val Trp Glu Ala Gly Xaa Thr Ala Asn Glu Ser Cys Pro Val Gly Ser
100 105 110
Xaa Arg Xaa Xaa Ser Xaa Xaa Xaa Ser Cys His Gln Gly Arg Ile Pro
115 120 125
Leu Tyr Ser Ala Ala Val Glu Ser Thr Xaa Gln Val Gln Xaa Ala Val
130 135 140
Arg Phe Ala Arg Arg His Asn Leu Arg Leu Val Ile Arg Asn Thr Gly
145 150 155 160
His Asp Xaa Ala Gly Arg Ser Ser Ala Pro Asp Ser Phe Gln Ile His
165 170 175
Thr His Arg Leu Gln Glu Ile Xaa Phe His Xaa Asn Phe Arg Leu Xaa
180 185 190
Gly Ser Xaa Xaa Xaa Xaa Thr Ser Leu Gly Xaa Pro Ala Val Thr Val
195 200 205
Gly Ala Gly Val Met Met Gly Glu Leu Tyr Ala Xaa Gly Ala Arg Xaa
210 215 220
Gly Tyr Met Val Leu Gly Gly Glu Cys Pro Thr Val Gly Val Val Gly
225 230 235 240
Gly Phe Leu Gln Gly Gly Gly Val Ser Ser Phe Leu Ser Phe Xaa Xaa
245 250 255
Gly Leu Ala Val Asp Asn Val Leu Glu Tyr Glu Val Val Thr Ala Xaa
260 265 270
Gly Glu Leu Val Val Ala Asn Ala His Gln Asn Gln Asp Leu Phe Trp
275 280 285
Ala Leu Arg Gly Gly Gly Gly Gly Thr Phe Gly Val Val Thr Arg Ala
290 295 300
Thr Met Arg Val Phe Pro Asp Val Pro Val Val Val Ser Glu Ile Leu
305 310 315 320
Leu Xaa Ala Xaa Xaa Ala Xaa Ser Ser Phe Trp Thr Xaa Gly Leu Ser
325 330 335
Xaa Leu Leu Thr Ala Leu Gln Ser Leu Asn Gln Asp Asn Val Gly Gly
340 345 350
Gln Leu Val Ile Xaa Xaa Xaa Xaa Lys Asp Ala Val Gln Ala Ser Ile
355 360 365
Xaa Leu Phe Phe Leu Asn Met Thr Glu Xaa Ala Xaa Ile Asp Xaa Arg
370 375 380
Met Lys Pro Xaa Leu Thr Xaa Leu Ser Xaa Xaa Asn Ile Xaa Tyr Thr
385 390 395 400
Xaa Ser Ser Lys Xaa Leu Pro Xaa Xaa Ser Ser Xaa Leu Arg Xaa Xaa
405 410 415
Pro Asp Ile His Pro Asp Asn Xaa Tyr Gly Ile Leu Xaa Ser Ser Val
420 425 430
Leu Ile Ser Gln Xaa Leu Phe Xaa Ser Ser Glu Gly Pro Xaa Lys Val
435 440 445
Ala Xaa Xaa Leu Ala Xaa Leu Pro Met Ser Pro Xaa Asp Leu Leu Phe
450 455 460
Thr Ser Asn Leu Gly Gly Arg Val Ile Ala Asn Gly Xaa Xaa Glu Leu
465 470 475 480
Ala Glu Thr Ser Met His Pro Ala Trp Arg Ser Ala Ala Gln Leu Ile
485 490 495
Asn Tyr Val Arg Thr Val Xaa Glu Pro Ser Ile Glu Gly Lys Ala Xaa
500 505 510
Ala Leu Glu Xaa Leu Thr Asn Xaa Gln Met Pro Ile Leu Tyr Ala Ile
515 520 525
Asp Pro Xaa Phe Lys Leu Ser Tyr Arg Asn Leu Gly Asp Pro Asn Glu
530 535 540
Lys Asp Phe Gln Gln Val Tyr Trp Gly Xaa Asn Xaa Tyr Gly Arg Leu
545 550 555 560
Ser Xaa Ile Lys Lys Lys Trp Asp Pro Asp Asp Leu Phe Phe Ser Lys
565 570 575
Leu Gly Val Gly Ser Glu Xaa Trp Asp Ser Glu Gly Met Cys Arg Lys
580 585 590
Xaa Xaa Xaa Xaa Gln Xaa Xaa Leu Xaa Gln Xaa Leu Xaa Xaa Leu Xaa
595 600 605
Ser
<210> 154
<211> 344
<212> PRT
<213> Artificial Sequence
<220>
<223> EasF consensus
<220>
<221> misc_feature
<222> (2)..(3)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (5)..(5)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (11)..(11)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (13)..(13)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (24)..(24)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (27)..(27)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (29)..(29)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (45)..(45)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (64)..(64)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (68)..(68)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (71)..(71)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (79)..(79)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (105)..(105)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (107)..(107)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (121)..(121)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (125)..(125)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (130)..(130)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (132)..(132)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (150)..(150)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (153)..(153)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (189)..(189)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (193)..(193)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (195)..(195)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (197)..(198)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (240)..(241)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (243)..(254)
<223> the stretch of X between amino acid position 243 and 254 can be
between 4 X and 12 X
<220>
<221> misc_feature
<222> (286)..(286)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (293)..(293)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (312)..(313)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (316)..(316)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (320)..(320)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (328)..(329)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (332)..(332)
<223> Xaa can be any naturally occurring amino acid
<400> 154
Met Xaa Xaa Pro Xaa Val Ile Asp Ile Arg Xaa Ser Xaa Phe Glu Asp
1 5 10 15
Ser Ile Pro Asp Gln Val Ile Xaa Gly Leu Xaa Ser Xaa Pro Lys Thr
20 25 30
Leu Pro Ala Leu Leu Phe Tyr Ser Asn Glu Gly Leu Xaa His Trp Asn
35 40 45
His His Ser His Gln Pro Asp Phe Tyr Pro Arg His Gln Glu Ile Xaa
50 55 60
Ile Leu Lys Xaa Lys Ala Xaa Glu Met Ala Ala Ser Ile Ala Xaa Asn
65 70 75 80
Ser Val Val Val Asp Leu Gly Ser Ala Ser Leu Asp Lys Val Ile His
85 90 95
Leu Leu Glu Ala Leu Glu Ala Gln Xaa Lys Xaa Val Thr Tyr Tyr Ala
100 105 110
Leu Asp Leu Ser Ala Ser Gln Leu Xaa Ser Thr Leu Xaa Ala Ile Pro
115 120 125
Thr Xaa Gln Xaa Phe Arg His Val Arg Phe Ala Gly Leu His Gly Thr
130 135 140
Phe Asp Asp Gly Leu Xaa Trp Leu Xaa Glu Thr Pro Glu Ile Arg Asp
145 150 155 160
Leu Pro His Cys Val Leu Leu Phe Gly Leu Thr Ile Gly Asn Phe Ser
165 170 175
Arg Pro Asn Ala Ala Ala Phe Leu Arg Asn Ile Ala Xaa His Ala Leu
180 185 190
Xaa Gly Xaa Ser Xaa Xaa Gln Ser Ser Ile Leu Leu Ser Leu Asp Ser
195 200 205
Cys Lys Val Pro Thr Gln Val Leu Arg Ala Tyr Thr Ala Glu Gly Val
210 215 220
Val Pro Phe Ala Leu Gln Ser Leu Thr Tyr Ala Asn Thr Leu Phe Xaa
225 230 235 240
Xaa Lys Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Val Phe
245 250 255
Asn Pro Asp Asp Trp Tyr Tyr Leu Ser Glu Trp Asn Phe Val Leu Gly
260 265 270
Arg His Glu Ala Ser Leu Ile Pro Arg Ser Lys Asp Ile Xaa Leu Gly
275 280 285
Ala Pro Leu Asp Xaa Ile Val Val Arg Lys Asp Glu Lys Val Arg Phe
290 295 300
Gly Cys Ser Tyr Lys Tyr Asp Xaa Xaa Glu Arg Xaa Glu Leu Phe Xaa
305 310 315 320
Ala Ala Gly Leu Lys Asp Glu Xaa Xaa Trp Ser Xaa Glu Gly Cys Asp
325 330 335
Val Ala Phe Tyr Gln Leu Lys Leu
340
<210> 155
<211> 318
<212> PRT
<213> Artificial Sequence
<220>
<223> EasG consensus
<220>
<221> misc_feature
<222> (21)..(22)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (24)..(26)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (38)..(41)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (46)..(48)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (55)..(55)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (60)..(60)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (63)..(64)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (66)..(70)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (73)..(73)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (82)..(82)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (86)..(86)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (124)..(124)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (133)..(133)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (137)..(137)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (139)..(139)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (152)..(154)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (160)..(160)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (163)..(163)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (182)..(182)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (196)..(196)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (198)..(198)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (201)..(201)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (217)..(235)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (259)..(259)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (262)..(263)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (265)..(265)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (269)..(269)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (273)..(273)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (281)..(281)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (284)..(284)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (292)..(294)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (297)..(297)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (301)..(302)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (312)..(312)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (315)..(316)
<223> Xaa can be any naturally occurring amino acid
<400> 155
Met Thr Ile Leu Leu Thr Gly Gly Arg Gly Lys Thr Ala Ser His Ile
1 5 10 15
Ala Ser Leu Leu Xaa Xaa Ala Xaa Xaa Xaa Val Pro Phe Ile Val Ala
20 25 30
Ser Arg Ser Ser Ser Xaa Xaa Xaa Xaa Ser Pro Tyr Arg Xaa Xaa Xaa
35 40 45
Phe Asp Trp Leu Asp Glu Xaa Thr Tyr Gly Asn Xaa Leu Ser Xaa Xaa
50 55 60
Asp Xaa Xaa Xaa Xaa Xaa Gly Met Xaa Pro Ile Ser Ala Val Trp Leu
65 70 75 80
Val Xaa Pro Pro Ile Xaa Asp Leu Ala Pro Pro Met Ile Lys Phe Ile
85 90 95
Asp Phe Ala Arg Ser Lys Gly Val Lys Arg Phe Val Leu Leu Ser Ala
100 105 110
Ser Thr Ile Glu Lys Gly Gly Pro Ala Met Gly Xaa Ile His Ala His
115 120 125
Leu Asp Ser Leu Xaa Glu Gly Ile Xaa Tyr Xaa Val Leu Arg Pro Thr
130 135 140
Trp Phe Met Glu Asn Phe Ser Xaa Xaa Xaa Glu Leu Gln Trp Ile Xaa
145 150 155 160
Ile Arg Xaa Glu Asn Lys Ile Tyr Ser Ala Thr Gly Asp Gly Lys Ile
165 170 175
Pro Phe Ile Ser Val Xaa Asp Ile Ala Arg Val Ala Phe Arg Ala Leu
180 185 190
Thr Asp Glu Xaa Ser Xaa Asn Thr Xaa Asp Tyr Val Leu Leu Gly Pro
195 200 205
Glu Leu Leu Thr Tyr Asp Asp Val Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
210 215 220
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Ala Glu Ile Leu Ser
225 230 235 240
Thr Val Leu Gly Arg Lys Ile Thr His Val Lys Leu Thr Glu Ala Glu
245 250 255
Leu Ala Xaa Lys Leu Xaa Xaa Glu Xaa Gly Met Pro Xaa Asp Asp Ala
260 265 270
Xaa Met Leu Ala Ser Met Asp Thr Xaa Val Lys Xaa Gly Ala Glu Glu
275 280 285
Arg Leu Asn Xaa Xaa Xaa Val Lys Xaa Val Thr Gly Xaa Xaa Pro Arg
290 295 300
Thr Phe Leu Asp Phe Ala Ser Xaa Glu Lys Xaa Xaa Trp Leu
305 310 315
<210> 156
<211> 285
<212> PRT
<213> Artificial Sequence
<220>
<223> EasH consensus
<220>
<221> misc_feature
<222> (3)..(3)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (5)..(5)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (10)..(10)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (14)..(14)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (16)..(16)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (20)..(21)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (33)..(34)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (39)..(42)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (45)..(46)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (49)..(49)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (51)..(51)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (55)..(55)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (57)..(57)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (63)..(63)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (65)..(65)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (68)..(68)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (72)..(72)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (74)..(74)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (85)..(85)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (89)..(89)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (93)..(94)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (99)..(99)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (102)..(103)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (107)..(107)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (122)..(122)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (126)..(126)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (129)..(129)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (146)..(146)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (150)..(150)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (163)..(163)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (180)..(180)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (182)..(187)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (189)..(189)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (194)..(194)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (204)..(204)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (215)..(217)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (242)..(242)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (245)..(245)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (262)..(262)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (269)..(269)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (280)..(280)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (282)..(283)
<223> Xaa can be any naturally occurring amino acid
<400> 156
Met Thr Xaa Thr Xaa Thr Ser Lys Pro Xaa Leu Arg Arg Xaa Pro Xaa
1 5 10 15
Ser Ala Gly Xaa Xaa Ala Ile Phe Gln Val Leu Gln Glu Asp Gly Val
20 25 30
Xaa Xaa Ile Glu Gly Phe Xaa Xaa Xaa Xaa Gln Val Xaa Xaa Phe Asn
35 40 45
Xaa Glu Xaa Asp Pro His Xaa Lys Xaa Trp Glu Leu Gly Gln Xaa Ser
50 55 60
Xaa Gln Glu Xaa Tyr Leu Ala Xaa Met Xaa Gln Leu Ser Ser Leu Pro
65 70 75 80
Leu Phe Ser Lys Xaa Phe Arg Asp Xaa Leu Met Asn Xaa Xaa Leu Leu
85 90 95
His Gly Xaa Cys Lys Xaa Xaa Phe Gly Pro Xaa Ser Gly Asp Tyr Trp
100 105 110
Leu Thr Thr Ser Ser Val Leu Glu Thr Xaa Pro Gly Tyr Xaa Gly Gln
115 120 125
Xaa Leu His Arg Glu His Asp Gly Ile Pro Ile Cys Thr Thr Leu Gly
130 135 140
Arg Xaa Ser Pro Glu Xaa Met Leu Asn Phe Leu Thr Ala Leu Thr Asp
145 150 155 160
Phe Thr Xaa Glu Asn Gly Ala Thr Arg Val Leu Pro Gly Ser His Leu
165 170 175
Trp Glu Asp Xaa Ser Xaa Xaa Xaa Xaa Xaa Xaa Asp Xaa Ala Ile Pro
180 185 190
Ala Xaa Met Asn Pro Gly Asp Ala Val Leu Phe Xaa Gly Lys Thr Leu
195 200 205
His Gly Ala Gly Lys Asn Xaa Xaa Xaa Asp Phe Leu Arg Arg Gly Phe
210 215 220
Pro Leu Ile Met Gln Ser Cys Gln Phe Thr Pro Val Glu Ala Ser Val
225 230 235 240
Ala Xaa Pro Arg Xaa Leu Val Glu Thr Met Thr Pro Leu Ala Gln Lys
245 250 255
Met Val Gly Trp Arg Xaa Val Ser Ala Lys Gly Val Xaa Ile Trp Thr
260 265 270
Tyr Asp Leu Lys Asp Leu Ala Xaa Gly Xaa Xaa Leu Lys
275 280 285
<210> 157
<211> 870
<212> DNA
<213> Artificial Sequence
<220>
<223> EasH of Paecilomyces divaricatus codon optimized for S.
cerrevisiae
<400> 157
atgaccgtga ccaaaacttc caagcccgtc cttcggcgct tgccgatctc cgccggagcc 60
gatgccattt tccaggttct ccaggaggac ggcgtcctca tcatcgaggg cttcctgacg 120
cccgagcagg tgaagaagtt taacgcggag gtcgaccccc acctcaagaa gtgggagttg 180
ggtcagggtt ccatgcaaga gggctacctc gccaacatga agcagctgtc cagcttgcct 240
ctcttcagca agaccttccg cgacgacctt atgaaccatg acctcctcca cggcatctgc 300
aagaaggcct tcggtccgga ttctggtgac tactggctga ctacgtcctc cgtcctggag 360
accgaccctg gctatcccgg ccaagacttg catcgggaac acgatggcat ccccatctgc 420
accaccctgg gccgtgactc ccctgaggcc atgctgaact tcctgaccgc tctgaccgac 480
tttaccgaag agaacggtgc cactcgcgtc ctgcccggtt cccacctgtg ggaggattac 540
tctaagagcg ttagtcccga ccaggcaatt cctgccgaga tgaatccggg tgacgccgtc 600
ttgttcagcg gcaagaccct ccacggtgcc ggcaagaacc agtccaagga cttcctccgc 660
cgtggcttcc ccctcattat gcagtcgtgt cagttcactc ccgtcgaggc cagcgtggct 720
atccctcgct ccctggtcga gaccatgacc cccctggctc agaagatggt cggctggcgt 780
tctgtctcgg ccaaaggagt ggaaatctgg acatacgacc tcaaggatct ggctaaggga 840
cttaagctga aggcgatcgc tgctgagtag 870
<210> 158
<211> 867
<212> DNA
<213> Saccharomyces cerevisiae
<400> 158
atgactgccg acaacaatag tatgccccat ggtgcagtat ctagttacgc caaattagtg 60
caaaaccaaa cacctgaaga cattttggaa gagtttcctg aaattattcc attacaacaa 120
agacctaata cccgatctag tgagacgtca aatgacgaaa gcggagaaac atgtttttct 180
ggtcatgatg aggagcaaat taagttaatg aatgaaaatt gtattgtttt ggattgggac 240
gataatgcta ttggtgccgg taccaagaaa gtttgtcatt taatggaaaa tattgaaaag 300
ggtttactac atcgtgcatt ctccgtcttt attttcaatg aacaaggtga attactttta 360
caacaaagag ccactgaaaa aataactttc cctgatcttt ggactaacac atgctgctct 420
catccactat gtattgatga cgaattaggt ttgaagggta agctagacga taagattaag 480
ggcgctatta ctgcggcggt gagaaaacta gatcatgaat taggtattcc agaagatgaa 540
actaagacaa ggggtaagtt tcacttttta aacagaatcc attacatggc accaagcaat 600
gaaccatggg gtgaacatga aattgattac atcctatttt ataagatcaa cgctaaagaa 660
aacttgactg tcaacccaaa cgtcaatgaa gttagagact tcaaatgggt ttcaccaaat 720
gatttgaaaa ctatgtttgc tgacccaagt tacaagttta cgccttggtt taagattatt 780
tgcgagaatt acttattcaa ctggtgggag caattagatg acctttctga agtggaaaat 840
gacaggcaaa ttcatagaat gctataa 867
<210> 159
<211> 1658
<212> DNA
<213> Artificial Sequence
<220>
<223> T/P cassette including both Asc I sites
<400> 159
ggcgcgccat gcagggatat cagatctggg ccgtatactt acatatagta gatgtcaagc 60
gtaggcgctt cccctgccgg ctgtgagggc gccataacca aggtatctat agaccgccaa 120
tcagcaaact acctccgtac attcatgttg cacccacaca tttatacacc cagaccgcga 180
caaattaccc ataaggttgt ttgtgacggc gtcgtacaag agaacgtggg aactttttag 240
gctcaccaaa aaagaaagaa aaaatacgag ttgctgacag aagcctcaag aaaaaaaaaa 300
ttcttcttcg actatgctgg aggcagagat gatcgagccg gtagttaact atatatagct 360
aaattggttc catcaccttc ttttctggtg tcgctccttc tagtgctatt tctggctttt 420
cctatttttt tttttccatt tttctttctc tctttctaat atataaattc tcttgcattt 480
tctatttttc tctctatcta ttctacttgt ttattccctt caaggttttt ttttaaggag 540
tacttgtttt tagaatatac ggtcaacgaa ctataattaa ctaaacaagc ttggcctgca 600
gggccagctt acccttaaat ttatttgcac tactggaaaa ctacctgttc catggccaac 660
acttgtcact actttctctt atggtgttca atgcttttcc cgttatccgg atcatatgaa 720
acggcatgac tttttcaaga gtgccatgcc cgaaggttat gtacaggaac gcactatatc 780
tttcaaagat gacgggaact acaagacgcg tgctgaagtc aagtttgaag gtgataccct 840
tgttaatcgt atcgagttaa aaggtattga ttttaaagaa gatggaaaca ttctcggaca 900
caaactcgag tacaactata actcacacaa tgtatacatc acggcagaca aacaaaagaa 960
tggaatcaaa gctaacttca aaattcgcca caacattgaa gatggatccg ttcaactagc 1020
agaccattat caacaaaata ctccaattgg cgatggccct gtccttttac cagacaacca 1080
ttacctgtcg acacaatctg ccctttcgaa agatcccaac gaaaagcgtg accacatggt 1140
ccttcttgag tttgtaactg ctgctgggat tacacatggc atggatgagc tctacaaata 1200
atgaattcgt cgaggccgtt caggcctcga ggccgttcag gctcgacccg gggatccgcg 1260
gacaaatcgc tcttaaatat atacctaaag aacattaaag ctatattata agcaaagata 1320
cgtaaatttt gcttatatta ttatacacat atcatatttc tatattttta agatttggtt 1380
atataatgta cgtaatgcaa aggaaataaa ttttatacat tattgaacag cgtccaagta 1440
actacattat gtgcactaat agtttagcgt cgtgaagact ttattgtgtc gcgaaaagta 1500
aaaattttaa aaattagagc accttgaact tgcgaaaaag gttctcatca actgtttaaa 1560
aggaggatat caggtcctat ttctgacaaa caatatacaa atttagtttc aaagcatgcc 1620
catgggttaa ctgatcaatg catcctgcat ggcgcgcc 1658
<210> 160
<211> 7599
<212> DNA
<213> Artificial Sequence
<220>
<223> pEVE2656 (YERC?8-KanMX-G/C+Aj_EasC)
<400> 160
tcgcgcgttt cggtgatgac ggtgaaaacc tctgacacat gcagctcccg gagacggtca 60
cagcttgtct gtaagcggat gccgggagca gacaagcccg tcagggcgcg tcagcgggtg 120
ttggcgggtg tcggggctgg cttaactatg cggcatcaga gcagattgta ctgagagtgc 180
accatatgcg gtgtgaaata ccgcacagat gcgtaaggag aaaataccgc atcaggcgcc 240
attcgccatt caggctgcgc aactgttggg aagggcgatc ggtgcgggcc tcttcgctat 300
tacgccagct ggcgaaaggg ggatgtgctg caaggcgatt aagttgggta acgccagggt 360
tttcccagtc acgacgttgt aaaacgacgg ccagtgaatt cgagctccct gcaggcccag 420
ttgtttgtag ctggttcata tttagcggca attctctgtt gcgtaaatga aaatattaat 480
gtaaacaaaa aaagaccaaa acattttagc agtgtaagaa ggtgtactga tacaaaatgt 540
gtttagagtc tactgatatg ttactgaccg ttcgttggga aaaaaatact gtatcattta 600
ttaatcaaaa gcgacttttg gtggaatatt atgatatgtg ttgttaaaat atgacgtaat 660
tttagaattg tctgattcgt attcaaattt ggtgaaggaa taacgcagag ttgacaattt 720
aatagaatgg attaatcgta attttcagaa acgtagaaaa agaaaaacaa ttaaaacatt 780
atattaagat tattgatttg ccttttaagg gtccagcggc cgccagctga agcttcgtac 840
gctgcaggtc gacaaccctt aatataactt cgtataatgt atgctatacg aagttattag 900
gtctagagat ctgtttagct tgcctcgtcc ccgccgggtc acccggccag cgacatggag 960
gcccagaata ccctccttga cagtcttgac gtgcgcagct caggggcatg atgtgactgt 1020
cgcccgtaca tttagcccat acatccccat gtataatcat ttgcatccat acattttgat 1080
ggccgcacgg cgcgaagcaa aaattacggc tcctcgctgc agacctgcga gcagggaaac 1140
gctcccctca cagacgcgtt gaattgtccc cacgccgcgc ccctgtagag aaatataaaa 1200
ggttaggatt tgccactgag gttcttcttt catatacttc cttttaaaat cttgctagga 1260
tacagttctc acatcacatc cgaacataaa caaccatggg taaggaaaag actcacgttt 1320
cgaggccgcg attaaattcc aacatggatg ctgatttata tgggtataaa tgggctcgcg 1380
ataatgtcgg gcaatcaggt gcgacaatct atcgattgta tgggaagccc gatgcgccag 1440
agttgtttct gaaacatggc aaaggtagcg ttgccaatga tgttacagat gagatggtca 1500
gactaaactg gctgacggaa tttatgcctc ttccgaccat caagcatttt atccgtactc 1560
ctgatgatgc atggttactc accactgcga tccccggcaa aacagcattc caggtattag 1620
aagaatatcc tgattcaggt gaaaatattg ttgatgcgct ggcagtgttc ctgcgccggt 1680
tgcattcgat tcctgtttgt aattgtcctt ttaacagcga tcgcgtattt cgtctcgctc 1740
aggcgcaatc acgaatgaat aacggtttgg ttgatgcgag tgattttgat gacgagcgta 1800
atggctggcc tgttgaacaa gtctggaaag aaatgcataa gcttttgcca ttctcaccgg 1860
attcagtcgt cactcatggt gatttctcac ttgataacct tatttttgac gaggggaaat 1920
taataggttg tattgatgtt ggacgagtcg gaatcgcaga ccgataccag gatcttgcca 1980
tcctatggaa ctgcctcggt gagttttctc cttcattaca gaaacggctt tttcaaaaat 2040
atggtattga taatcctgat atgaataaat tgcagtttca tttgatgctc gatgagtttt 2100
tctaatcagt actgacaata aaaagattct tgttttcaag aacttgtcat ttgtatagtt 2160
tttttatatt gtagttgttc tattttaatc aaatgttagc gtgatttata ttttttttcg 2220
cctcgacatc atctgcccag atgcgaagtt aagtgcgcag aaagtaatat catgcgtcaa 2280
tcgtatgtga atgctggtcg ctatactgct gtcgattcga tactaacgcc gccatccagt 2340
gtcgaaaacg agctctcgag aacccttaat ataacttcgt ataatgtatg ctatacgaag 2400
ttattaggtg atatcagatc cactagtggc ctatgcggcc gcggcgcgcc agcttgcaaa 2460
ttaaagcctt cgagcgtccc aaaaccttct caagcaaggt tttcagtata atgttacatg 2520
cgtacacgcg cccagttcga gtttatcatt atcaatactg ccatttcaaa gaatacgtaa 2580
ataattaata gtagtgattt tcctaacttt atttagtcaa aaaattagcc ttttaattct 2640
gctgtaaccc gtacatgccc aaaatagggg gcgggttaca cagaatatat aacatcgtag 2700
gtgtctgggt gaacagttta ttcctggcat ccactaaata taatggagcc cgctttttaa 2760
gctggcatcc agaaaaaaaa agaatcccag caccaaaata ttgttttctt caccaaccat 2820
cagttcatag gtccattctc ttagcgcaac tacagagaac aggggcacaa acaggcaaaa 2880
aacggacaca acctcaatgg agtgatgcaa cctgcctgga gtaaatgatg acacaaggca 2940
attgacccac gcatgtatct atctcatttt cttacacctt ctattacctt ctgctctctc 3000
tgatttggaa aaagctgaaa aaaaaggttg aaaccagttc cctgaaatta ttcccctact 3060
tgactaataa gtatataaag acggtaggta ttgattgtaa ttctgtaaat ctatttctta 3120
aacttcttaa attctacttt tatagttagt ctttttttta gttttaaaac accaagaact 3180
tagtttcgaa taaacacaca taaacaaaca aaaagcttaa aatggctcca gattctaagc 3240
caacttacac tactgctaat ggttgtccat ttcataagcc tgaaggtcca ccagctgatg 3300
gtagaacttt ggctttgtct gatcatcatt tggttgatac cttggcccat ttcaacagag 3360
aaaagattcc agaaagagtt gttcacgcta aaggtgctgg tgcttatggt gaatttgaag 3420
ttactgccga tatctccgat atttgcgata tcgatatgtt ggttggtgtc ggtaaaaaga 3480
ctccatgtgt tactagattc tctaccactg gtttggaaag aggttctaac gaaggtatga 3540
gagatttgaa aggtatggct tgtaagttct acactactga aggtaattgg gattgggtca 3600
tgttgaattt cccattcttc ttcattcgtg atccagtcaa gttcccatct ttgatgcacg 3660
ctcaaagaag agatccacaa actaatttgt tgaacccaaa catgtactgg gattgggtta 3720
ccaacaatca tgaatccttg cacatggtct tgttgcaatt ttctgatttc ggtactatgt 3780
tcaattggag atccatgtct ggttatatgg ctcatgctta caaatgggtt aagccagatg 3840
gttcttttaa gtacgtccac atcttcttgt catctgatag aggtccaaat ttcaccgatg 3900
gtcaacaagc taaaggtaca aatgatttgg atccagatca cgctactaga gacttgtatg 3960
aagctattga aagaggtgaa tacccaactt ggactgcttc tgttcaagtt gttgatccaa 4020
aagatgctcc aaacttgggt tacaacattt tggatgttac taagcactgg aacttgggta 4080
cttacccaaa agatgttact ttgatcccac caaaggtttt cggtaagttg actttgaaaa 4140
gaaacccagc taactacttc gccgaaattg aacaattggc ttactctcca tctaacatgg 4200
ttccaggtgt tgctccatct gaagatccaa ttttacaagc tagaattttc gcttacccag 4260
acgctcaaag atatagattg ggtgctaacc atcaacaaat cccagttaat agatccgctc 4320
atacctttaa cccaattgct agagatggtc aaggtacttt tgatgctaat tatggtgctc 4380
atccaggttt cttgactcaa caacaaccag ttagatttgc cgaacctaga gaaccagatc 4440
caaagtataa tgaatggttg aaagaaatcc aatccaagtc ctggttgcaa actactgaac 4500
atgattacaa gttcgccaga gatttctacg aagttttgcc agattttaga ggtcaagaat 4560
tccaagatac catggtccaa aacatggttg aatctgttgc tcaaaccaga gccgaaattc 4620
aaaagcaagt ttacgaaacc tggaagttgg tttctccagc tttggctgct agaattcaaa 4680
aaggtgttga aaccttgttg caaaagtctg aagctccagc tgaagaatct tttattccag 4740
ctagattgtg accgcggcac gtccgacggc ggcccgacgg gtccgaggcc tcggagatcc 4800
gtcccccttt tcctttgtcg atatcatgta attagttatg tcacgcttac attcacgccc 4860
tccccccaca tccgctctaa ccgaaaagga aggagttaga caacctgaag tctaggtccc 4920
tatttatttt tttatagtta tgttagtatt aagaacgtta tttatatttc aaatttttct 4980
tttttttctg tacagacgcg cccgatggat gatgattggt tcttatcata atttgatttc 5040
ggcagaagca atattagagg tattgttgta acgaaattcc aatgtcatct gcttagtatt 5100
attaatgtta cctgaatatt atcacatgct gcttaaaaat gtgttataag tattaaaatc 5160
tagtgaaagt tgaaatgtaa tctaatagga taatgaaaca tatgaaacgg aatgaggaat 5220
aatcgttgta tcactatgta gagatatcga tttcattttg aggattccta tattcttggg 5280
gagaacttct actatattct gtatacatga tataatagcc tctcccaaca atggaatgcc 5340
aacaacctgc agggcatgca agcttggcgt aatcatggtc atagctgttt cctgtgtgaa 5400
attgttatcc gctcacaatt ccacacaaca tacgagccgg aagcataaag tgtaaagcct 5460
ggggtgccta atgagtgagc taactcacat taattgcgtt gcgctcactg cccgctttcc 5520
agtcgggaaa cctgtcgtgc cagctgcatt aatgaatcgg ccaacgcgcg gggagaggcg 5580
gtttgcgtat tgggcgctct tccgcttcct cgctcactga ctcgctgcgc tcggtcgttc 5640
ggctgcggcg agcggtatca gctcactcaa aggcggtaat acggttatcc acagaatcag 5700
gggataacgc aggaaagaac atgtgagcaa aaggccagca aaaggccagg aaccgtaaaa 5760
aggccgcgtt gctggcgttt ttccataggc tccgcccccc tgacgagcat cacaaaaatc 5820
gacgctcaag tcagaggtgg cgaaacccga caggactata aagataccag gcgtttcccc 5880
ctggaagctc cctcgtgcgc tctcctgttc cgaccctgcc gcttaccgga tacctgtccg 5940
cctttctccc ttcgggaagc gtggcgcttt ctcatagctc acgctgtagg tatctcagtt 6000
cggtgtaggt cgttcgctcc aagctgggct gtgtgcacga accccccgtt cagcccgacc 6060
gctgcgcctt atccggtaac tatcgtcttg agtccaaccc ggtaagacac gacttatcgc 6120
cactggcagc agccactggt aacaggatta gcagagcgag gtatgtaggc ggtgctacag 6180
agttcttgaa gtggtggcct aactacggct acactagaag gacagtattt ggtatctgcg 6240
ctctgctgaa gccagttacc ttcggaaaaa gagttggtag ctcttgatcc ggcaaacaaa 6300
ccaccgctgg tagcggtggt ttttttgttt gcaagcagca gattacgcgc agaaaaaaag 6360
gatctcaaga agatcctttg atcttttcta cggggtctga cgctcagtgg aacgaaaact 6420
cacgttaagg gattttggtc atgagattat caaaaaggat cttcacctag atccttttaa 6480
attaaaaatg aagttttaaa tcaatctaaa gtatatatga gtaaacttgg tctgacagtt 6540
accaatgctt aatcagtgag gcacctatct cagcgatctg tctatttcgt tcatccatag 6600
ttgcctgact ccccgtcgtg tagataacta cgatacggga gggcttacca tctggcccca 6660
gtgctgcaat gataccgcga gacccacgct caccggctcc agatttatca gcaataaacc 6720
agccagccgg aagggccgag cgcagaagtg gtcctgcaac tttatccgcc tccatccagt 6780
ctattaattg ttgccgggaa gctagagtaa gtagttcgcc agttaatagt ttgcgcaacg 6840
ttgttgccat tgctacaggc atcgtggtgt cacgctcgtc gtttggtatg gcttcattca 6900
gctccggttc ccaacgatca aggcgagtta catgatcccc catgttgtgc aaaaaagcgg 6960
ttagctcctt cggtcctccg atcgttgtca gaagtaagtt ggccgcagtg ttatcactca 7020
tggttatggc agcactgcat aattctctta ctgtcatgcc atccgtaaga tgcttttctg 7080
tgactggtga gtactcaacc aagtcattct gagaatagtg tatgcggcga ccgagttgct 7140
cttgcccggc gtcaatacgg gataataccg cgccacatag cagaacttta aaagtgctca 7200
tcattggaaa acgttcttcg gggcgaaaac tctcaaggat cttaccgctg ttgagatcca 7260
gttcgatgta acccactcgt gcacccaact gatcttcagc atcttttact ttcaccagcg 7320
tttctgggtg agcaaaaaca ggaaggcaaa atgccgcaaa aaagggaata agggcgacac 7380
ggaaatgttg aatactcata ctcttccttt ttcaatatta ttgaagcatt tatcagggtt 7440
attgtctcat gagcggatac atatttgaat gtatttagaa aaataaacaa ataggggttc 7500
cgcgcacatt tccccgaaaa gtgccacctg acgtctaaga aaccattatt atcatgacat 7560
taacctataa aaataggcgt atcacgaggc cctttcgtc 7599
<210> 161
<211> 1818
<212> DNA
<213> Artificial Sequence
<220>
<223> EasE of Neotyphodium lolii codon optimized for S.cerrevisiae
<400> 161
atgagacact tcgttacttt cgttgtcggt tttttgttgt cctggggttt cttgtcatct 60
ttacaagatc caccatcttg tagatgtaga ccatgggaac catgttggcc agattctcat 120
caatggacat ctttgaacgc ttccattgac ggtaatttgg ttcatgttca accagttggt 180
tctgtttgtc atgatccaca ttatgatgct gttgcttgtg gtgatgtttt ggacttgtct 240
agaaattctg gttggagagc ttctaatcca gctacattgc aagattggat ttgggaaact 300
ggttctggtg ataacgaatc ttgtttgttg gtcagttctt ccgaaagacc acaagaaatt 360
ccatgtcatc aaggtagatt gccattatat tctgctgctg ttaagtctac tgcacacgtt 420
caaggtgtta ttagattcgc taaggatcac aacttgagat tggttattaa gaacaccggt 480
catgatgcta ctggtagatc agctgctcca gattctttac aaattcacac ctactttttg 540
aaggacatcc actacgatga caatttctta gttcatggtg atgctacagg ttctggtcca 600
gctgttactt tgggtgctgg tgttgttcat tctgaagttt acaaacatgg tatcgaccac 660
aagtactctg ttgttggtgg tgaatgtcca actgttggta tagttggtgg ttttttacaa 720
ggtggtggtg tttcttcttg gtctggtttt actagaggtt tggctgttga taacgtcttg 780
gaataccaag ttgttactgc taacgctgaa ttggttatcg ctaacgaaca ccaaaatcaa 840
gatttgttct gggctttgag aggcggcggt ggtggtactt tcggtgttgt tactcaagct 900
actgttagag cttttccaga tgatcctact gttgtttcca ctttggtttt gtcatctact 960
agagctgata cttcattctg ggttaaggcc atctctagat tattgtccat tttgagatcc 1020
tgcaatcaac aaaacgttca cggtcaattg atcatcacta gaccatccgt tgatattttg 1080
aatgctggtt tgaccttgca cttctctaat atgaccaacg ttttacatgc cgaaaccttg 1140
ttgcaaccac atattgcttc attgtccgaa gatcaaattt ctaccacttt gacctctaag 1200
ttcgttgcca acattaactc cgaattgaga ttagatgccg atatccatcc aagaggtatc 1260
ggtactttac aaacctccat gatgatctcc aacgaattat tcggttcttc tgaaggtcca 1320
ttgactgttg ctcaagtttt tggtaaattg ccaatcggtc caaacgattt gttgttcaca 1380
tctaatttgg gtggttgcat tgctgctaac aaaggtttgg atactgctat tcatccagct 1440
tggagaagtt ctgctcattt ggttacttac gttagatccg ttgaaccatc cattgaagct 1500
aaaaagttgg ccttgaaaga aatcaccaac aagtacatgc caatcttgta ctctatgcaa 1560
ccatccttca aggtcagtta cagaaacttg ggtgatccaa acgaaaagaa ctaccaagaa 1620
gttttctggg gtgaaaaggt ctacaaaaga ttggcttcta ttaaggccaa gttggatcca 1680
gatggtttgt tcatctctaa attaggtgtc ggttctgaag attgggatac tgaaggtatg 1740
tgttaccaac cacaaaacag agtttctcaa ccattgagat cattgaagta ctttttgtcc 1800
gtcttgaaag atacttaa 1818
<210> 162
<211> 1866
<212> DNA
<213> Artificial Sequence
<220>
<223> EasE of A. japonicus codon optimized for S. cerrevisia Version 1
<400> 162
atgggccaat ctaggggcat tttaggtggt gtgagacaat tgatattagt cattttggtc 60
ggagcttatc tgtccagact atctgccgtc gatgacgaca gacatgattg tcgatgtcgt 120
cctggtgaac catgctggcc aacagtttca cattggtcta ccctcaacga aagcattggt 180
ggtgcacttg cacatgttaa accaatcgct catgtctgcc accaatctgg tcaggacagc 240
tctgcttgtg aacaagtttt acaagagagt ttagactcaa aatggagggc ttcccataca 300
ggtgcactgc aagattgggt ttgggaagga ggagcagaat ctaatcaaac ttgctactac 360
ctgcgtagtg gtcctgctgg ctcatgtcac caaggtagaa taccacttta cagtgcagct 420
gtcaagtctg cctccgatgt gcaaaaggtt gttgatttca ctagacagca taatttgaga 480
ttagtcatca gaaatacagg gcatgacgga tctggtagat catccggacc agactcagta 540
gagatacaca ctcaccattt gaactcagta caatatcatc caaactttag acctgcagga 600
agctcagaac gacagtctgc acctggtcaa cctgctgtga cagtcggcgc aggtatctta 660
cttggtgatc tctacgcgag aggggcctca gagggttgga ttgtagttgg aggcgaatgt 720
ccaactgttg gtgctgctgg tggatttttg caaggcggtg gggtatcttc atggctttcc 780
tacgcgcatg ggttagcggt ggataacgta ctagagtacg aagtggtaac agctaaagga 840
gaaattgtga tcgcaaatgc ccaccaaaat ccagacctat tctgggcatt gcgtggtgga 900
ggcggtggta catttggcgt tgtaacacaa gctacattac aagttcaccc tgaccttcca 960
gtgtctgtcg ccgacgccgt tgttacggga tctagagctg atgcgacatt ttggagtcat 1020
ggcgtggctg cattattgag agcgttacaa tttctaaaca atcatggtac tgcagggcag 1080
tttatcctta gaaataccga tgacgatact gtacaagcca gtctcacaat gtacttttct 1140
aacttgacag ttcctgccgt cgccgatgaa aggatggatc ctttgagaag agcattagag 1200
cataatggcc acccttacca actaacatcc agattcctac cacaaatctc ttccactttt 1260
agacacactg cagacagata ccctgaagat tatggtattt tgatgggatc tgttttggtt 1320
tcattggatt tgttcaattc agcaaccggt ccagccgctt tagctcaaca ttttgctaga 1380
ctgcctatga caccagatga cttgctgttc acttctaatc taggcggtag agttagttca 1440
cttaacagag atccagcttc tacggctatg cacccaggct ggagagatgc agctcagtta 1500
ctgaatttcg taagaggtgt gggtgctcca agtctagccg ccaaggctac tgcactacat 1560
gaacttcaaa ctgttcagat ggcgagattg tacgaaatcg aaccagcttt ccaaatcagt 1620
tacagaaacc ttggagatcc atctgagaga agatcaagag aagtatattg gggttctaac 1680
tatgcccgtt tggtcgaggt taaaaggaga tgggatccag aaggcttgtt tttctccaaa 1740
ctggggattg atggagatgc ctgggatgct gaagggatgt gtcgtcaaac cagacagggc 1800
gcttggaatg ttgcagtcaa gtgggttcaa tcatttctcg gatcattatc cgcatctgta 1860
gtgtaa 1866
<210> 163
<211> 1866
<212> DNA
<213> Artificial Sequence
<220>
<223> EasE of A. japonicus codon optimized for S. cerrevisiae Version 2
<400> 163
atgggccagt cgcggggtat cttgggtggc gtccgtcagc tcatcctcgt cattctggtg 60
ggcgcttatc tatctagact tagcgccgtg gacgacgacc gtcatgattg ccgctgtaga 120
cctggcgaac cctgctggcc gacagtgtca cattggtcca ccttgaatga gtctatcggc 180
ggagccctcg cgcacgttaa gcccattgcg cacgtctgcc accagtctgg ccaggacagt 240
tccgcttgtg agcaggttct gcaggagtct ctggatagca agtggcgagc ctcccacact 300
ggggctttgc aggattgggt gtgggaagga ggcgctgagt cgaaccagac ttgttactac 360
cttcgatccg gccctgctgg ctcctgccat cagggtcgaa tcccattgta cagcgctgcg 420
gtcaaaagcg ccagcgacgt tcagaaggtc gtcgacttca cccgccaaca caacctgcgt 480
ctggttattc ggaacaccgg acacgacggc agtgggcgga gcagtggtcc cgattcagtc 540
gagattcaca cccaccatct aaattctgtt cagtaccacc ccaacttccg tcccgccggt 600
agctccgaac gccaaagtgc acccggtcag cccgccgtta ctgtcggtgc cggaatcctg 660
ctgggtgatc tgtacgctcg cggcgcttcg gaaggatgga tcgttgtggg aggtgagtgc 720
cctactgtcg gtgctgcggg cggctttctg caaggcgggg gtgtcagctc gtggctttcc 780
tacgctcacg ggctcgcagt tgacaatgtg ctggaatatg aggtcgttac cgcgaagggc 840
gagatcgtca tcgctaacgc gcaccaaaat ccggatcttt tctgggcact ccgtggaggt 900
ggcggtggta cgtttggtgt ggtaacccag gctacactcc aagtgcatcc tgatctacca 960
gtgtccgtcg ctgatgcggt cgtcaccggc tcccgtgccg acgcaacctt ctggtcgcat 1020
ggcgtggcag ccctcttgcg cgccctgcag tttcttaaca accacgggac tgcgggccag 1080
ttcatcttac gcaacacgga cgatgatacc gtccaggcgt ccctgacaat gtacttctcg 1140
aacctgaccg taccggcagt tgcggacgag cgcatggatc cgctgcgtcg tgcactggag 1200
cataacggcc acccttacca gcttacgagc aggttcctcc cccaaattag ctccactttc 1260
cgtcatactg ccgacaggta ccccgaggac tacggcatct tgatgggcag tgtcctcgtc 1320
tccttggatt tgtttaattc tgctaccggt cctgccgctc tcgcacagca cttcgcccgc 1380
ctccccatga cgcctgacga tctccttttc acgtcgaacc tgggtggtcg agtgtcgtct 1440
ctcaaccggg acccagcttc caccgccatg caccccggtt ggcgcgatgc tgctcaattg 1500
cttaacttcg tccggggcgt gggtgccccg agcctcgccg ccaaggctac cgccctgcat 1560
gagctgcaga ccgttcagat ggctcgcttg tacgagatcg agccagcgtt ccagatcagc 1620
tatcgcaacc tcggagaccc gtcggagcgt cgctcccgag aggtctactg gggttccaac 1680
tatgcccggc tggtcgaagt caaacgcaga tgggaccccg agggactctt tttcagtaaa 1740
cttggcatag acggagatgc ctgggacgcc gaaggaatgt gccggcagac tcgtcaaggt 1800
gcttggaacg ttgccgtgaa gtgggtccaa tctttcctcg gctccctctc tgcctccgtg 1860
gtgtag 1866
<210> 164
<211> 1866
<212> DNA
<213> Artificial Sequence
<220>
<223> EasE of A. japonicus codon optimized for S. cerrevisiae Version 3
<400> 164
atgggccagt cccgtggcat cctgggtggc gttcgccagt tgatcctcgt gattctggtg 60
ggcgcctacc tttcccgcct ctccgccgtc gacgacgacc gccacgattg ccgctgccgt 120
cccggcgagc cttgctggcc taccgtctcc cactggtcca cgctgaacga gagcatcgga 180
ggtgccctcg ctcatgtcaa gcccatcgca cacgtgtgcc atcagtcggg ccaggactct 240
tccgcttgcg agcaggtgct tcaggagagc ctcgacagca agtggcgagc ctctcacacg 300
ggcgccctgc aggactgggt ctgggagggt ggcgccgagt ctaaccagac gtgctattat 360
ctgcgcagcg gtcctgcggg aagctgccac caaggccgta ttcccctgta ctctgccgct 420
gtcaagtccg cttccgacgt ccagaaagtc gtcgatttca cccgtcagca caacctccgc 480
ctcgtcatcc gtaacacggg tcacgatggt tctggccgct cgtccggccc cgactccgtc 540
gagatccata ctcaccatct caattccgtc cagtaccacc ctaacttccg tccggcgggt 600
agcagcgaac gtcagtccgc tcccggtcag cctgccgtca ccgtcggtgc cggcatcctg 660
ctgggcgact tgtacgctcg cggagccagc gagggctgga ttgtggtggg cggtgagtgt 720
cccaccgtgg gcgcggccgg cggtttcctt caaggcggcg gcgtctccag ctggctctct 780
tatgctcacg gcctggccgt cgacaacgtc ctcgaatacg aggtggtcac cgcgaagggt 840
gagatcgtca tcgccaacgc tcaccagaac cctgaccttt tctgggctct ccgtggagga 900
ggcggcggaa cctttggcgt tgtcacccag gccactctgc aggtccaccc cgatttgccc 960
gtgagtgtcg cggacgctgt ggttaccggc tcgcgcgcag acgccacctt ctggagccac 1020
ggtgtcgctg ccctcttgcg cgctttgcag ttcctcaaca accacggcac tgcgggtcag 1080
ttcatcctgc gtaacactga tgacgataca gtccaggcct ctctgaccat gtacttctcc 1140
aacctcaccg tccccgccgt ggccgacgag cgcatggacc ccctccgccg tgctctggag 1200
cacaacggac acccctacca attgactagc cgcttcctgc cccaaatttc ctccactttc 1260
cggcacaccg ccgaccgcta ccccgaggac tacggcatcc tgatgggatc ggtcctcgtc 1320
tccttggatc tgttcaactc ggccaccggc ccggccgctc tggcccagca cttcgcccgc 1380
cttcctatga cccccgacga cctgctgttt acttccaatc tgggcggtcg cgtcagcagc 1440
ctcaatcggg atccagccag taccgccatg caccccggct ggcgcgacgc tgcacagctt 1500
ctgaacttcg tccggggtgt tggcgccccc tccctcgccg cgaaggccac cgccctgcat 1560
gagttgcaga ccgttcagat ggctcggctc tacgagatcg agcctgcctt ccaaatctcg 1620
taccgtaacc tgggtgaccc gtccgagcgc cgttcccgtg aagtctactg gggctccaac 1680
tacgcccgcc tcgttgaagt gaagcggcgc tgggacccgg agggtctgtt tttctccaag 1740
ctcggcatcg acggtgatgc ctgggacgct gagggtatgt gtcggcagac ccgtcagggc 1800
gcctggaacg tcgctgttaa gtgggtgcag tccttcctgg gtagcctgtc tgcttctgtt 1860
gtgtag 1866
<210> 165
<211> 1866
<212> DNA
<213> Artificial Sequence
<220>
<223> EasE of A. japonicus codon optimized for S. cerrevisiae Version 4
<400> 165
atgggtcaat cccgaggtat tctcggtggt gttagacaat tgatcttggt tattttggtt 60
ggtgcctact tgtctagatt gtctgctgtt gatgatgata gacatgattg cagatgtaga 120
ccaggtgaac catgttggcc aactgtttct cattggtcta ctttgaacga atctattggt 180
ggtgctttgg ctcatgttaa gccaattgct catgtttgtc atcaatccgg tcaagattct 240
tctgcttgcg aacaagtttt acaagaatcc ttggattcta agtggcgtgc ttctcatact 300
ggtgctttac aagattgggt ttgggaaggt ggtgctgaat ctaatcaaac ttgttactac 360
ttgagatcag gtccagctgg ttcttgtcat caaggtagaa ttccattata ttccgctgct 420
gttaagtccg cttctgatgt tcaaaaggtt gttgatttca ccagacaaca caacttgaga 480
ttggttatta gaaacaccgg tcatgatggt tctggtagat catctggtcc agattctgtt 540
gaaattcata cccatcactt gaactccgtt caataccatc caaattttag accagctggt 600
tcctctgaaa gacaatctgc tccaggtcaa ccagctgtta ctgttggtgc tggtattttg 660
ttgggtgact tgtatgctag aggtgcttct gaaggttgga tagttgttgg tggtgaatgt 720
ccaacagttg gtgctgctgg tggtttttta caaggtggtg gtgtttcttc ttggttgtct 780
tatgctcatg gtctcgcggt tgataacgtc ctcgaatacg aagttgttac tgctaagggt 840
gaaatcgtta ttgctaacgc tcatcaaaac ccagatttgt tttgggcttt gagaggcggt 900
ggtggtggta cttttggtgt tgttactcaa gctacattgc aagttcatcc agatttgcca 960
gtttctgttg ctgatgctgt tgttactggt tctagagctg atgctacttt ttggtcacat 1020
ggtgttgctg ctttgttgag agctttacaa ttcttgaaca accatggtac tgccggtcaa 1080
ttcattttga gaaacactga tgatgatacc gtccaagcta gtttgactat gtacttctct 1140
aacttgactg ttccagctgt tgctgacgaa agaatggatc ctttgagaag agctttggaa 1200
cataatggtc atccatacca attgacctct agattcttgc cacaaatctc ttctactttc 1260
agacataccg ctgatagata cccagaagat tacggtattt tgatgggttc cgttttggtt 1320
tccttggact tgtttaattc tgctactggt ccagctgctt tggcacaaca ttttgctaga 1380
ttgccaatga ctccagatga tttgttgttc acttctaact tgggtggtag agtctcttca 1440
ttgaatagag atccagcttc tactgcgatg catccaggtt ggagagatgc tgctcaatta 1500
ttgaatttcg ttagaggtgt tggtgctcca tctttggctg ctaaagcgac tgcgttacat 1560
gaattgcaaa ccgttcaaat ggccagatta tacgaaattg aaccagcctt ccaaatctcc 1620
tacagaaatt tgggtgatcc atccgaaaga agatccagag aagtttattg gggttctaac 1680
tacgctagat tggttgaagt taagagaaga tgggatccag aaggtttgtt cttctccaaa 1740
ttgggtattg atggtgatgc ttgggatgct gaaggtatgt gtagacaaac aagacaaggt 1800
gcttggaatg ttgctgttaa gtgggttcaa tctttcttgg gttctttgtc tgcttccgtt 1860
gtttga 1866
<210> 166
<211> 1569
<212> DNA
<213> Saccharomyces cerevisiae
<400> 166
atgaagtttt ctgctggtgc cgtcctgtca tggtcctccc tgctgctcgc ctcctctgtt 60
ttcgcccaac aagaggctgt ggcccctgaa gactccgctg tcgttaagtt ggccaccgac 120
tccttcaatg agtacattca gtcgcacgac ttggtgcttg cggagttttt tgctccatgg 180
tgtggccact gtaagaacat ggctcctgaa tacgttaaag ccgccgagac tttagttgag 240
aaaaacatta ccttggccca gatcgactgt actgaaaacc aggatctgtg tatggaacac 300
aacattccag ggttcccaag cttgaagatt ttcaaaaaca gcgatgttaa caactcgatc 360
gattacgagg gacctagaac tgccgaggcc attgtccaat tcatgatcaa gcaaagccaa 420
ccggctgtcg ccgttgttgc tgatctacca gcttaccttg ctaacgagac ttttgtcact 480
ccagttatcg tccaatccgg taagattgac gccgacttca acgccacctt ttactccatg 540
gccaacaaac acttcaacga ctacgacttt gtctccgctg aaaacgcaga cgatgatttc 600
aagctttcta tttacttgcc ctccgccatg gacgagcctg tagtatacaa cggtaagaaa 660
gccgatatcg ctgacgctga tgtttttgaa aaatggttgc aagtggaagc cttgccctac 720
tttggtgaaa tcgacggttc cgttttcgcc caatacgtcg aaagcggttt gcctttgggt 780
tacttattct acaatgacga ggaagaattg gaagaataca agcctctctt taccgagttg 840
gccaaaaaga acagaggtct aatgaacttt gttagcatcg atgccagaaa attcggcaga 900
cacgccggca acttgaacat gaaggaacaa ttccctctat ttgccatcca cgacatgact 960
gaagacttga agtacggttt gcctcaactc tctgaagagg cgtttgacga attgagcgac 1020
aagatcgtgt tggagtctaa ggctattgaa tctttggtta aggacttctt gaaaggtgat 1080
gcctccccaa tcgtgaagtc ccaagagatc ttcgagaacc aagattcctc tgtcttccaa 1140
ttggtcggta agaaccatga cgaaatcgtc aacgacccaa agaaggacgt tcttgttttg 1200
tactatgccc catggtgtgg tcactgtaag agattggccc caacttacca agaactagct 1260
gatacctacg ccaacgccac atccgacgtt ttgattgcta aactagacca cactgaaaac 1320
gatgtcagag gcgtcgtaat tgaaggttac ccaacaatcg tcttataccc aggtggtaag 1380
aagtccgaat ctgttgtgta ccaaggttca agatccttgg actctttatt cgacttcatc 1440
aaggaaaacg gtcacttcga cgtcgacggt aaggccttgt acgaagaagc ccaggaaaaa 1500
gctgctgagg aagccgatgc tgacgctgaa ttggctgacg aagaagatgc cattcacgat 1560
gaattgtaa 1569
<210> 167
<211> 921
<212> DNA
<213> Saccharomyces cerevisiae
<400> 167
atgcagttga gcaaggctgc tgagatgtgt tatgagataa caaactctta cttacacata 60
gaccagaaat ctcagataat agcaagtaca caagaagcga tacggttgac aagaaaatac 120
ttactaagtg aaatttttgt acgttggagt ccactgaatg gggaaatatc attctcgtac 180
aacggaggaa aagattgcca ggtattacta ctgttatatc tgagttgctt atgggaatat 240
ttcttcatta aggctcaaaa ttcccaattc gatttcgagt ttcaaagctt ccccatgcaa 300
agacttccaa ctgttttcat tgatcaagaa gaaactttcc ctacattaga gaattttgta 360
ctggaaacct cagagcgata ttgcctttcc ttatacgaat cacaaaggca atctggtgca 420
tcggtcaata tggcagacgc atttagagat tttataaaga tataccctga gaccgaagct 480
atagtgatag gtattagaca cacagaccca tttggtgaag cattaaagcc tattcaaaga 540
acagattcta actggcctga ttttatgagg ttgcaacctc tcttacactg ggacttaacc 600
aatatatgga gtttcttact gtattctaat gagccaattt gtggactata tggtaaaggt 660
ttcacatcaa tcggcggaat taacaactca ttgcctaacc cacacttgag aaaggactcc 720
aataatccag ccttgcattt tgaatgggaa atcattcatg catttggcaa ggacgcagaa 780
ggcgaacgta gttccgctat aaacacgtca cctatttccg tggtggataa ggaaagattc 840
agcaaatacc atgacaatta ctatcctggc tggtatttgg ttgatgacac tttagagaga 900
gcaggcagga tcaagaatta a 921
<210> 168
<211> 1997
<212> DNA
<213> Artificial Sequence
<220>
<223> P/T cassette including both AscI sites
<400> 168
ggcgcgccat gcagggatat cagatctaat gctactattt tggagatcaa tctcagtaca 60
aaacaatatt aaaaagaggt gaattatttt tcccccctta tttttttttt gttagaattg 120
atccaaatgt aaataaacaa tcacaaggaa aaaaaaaaaa aaaaaaaata gccgccatga 180
ccccggatcg tcggttgtga tacggtcagg gtagcgccct ggtcaaactt cagaactaaa 240
aaaataataa ggaagaaaaa aatagctaat ttttccggca gaaagatttt cgctacccga 300
aagtttttcc ggcaagctaa atggaaaaag gaaagattat tgaaagagaa agaaagaaaa 360
aaaaaaaatg tacacccaga catcgggctt ccacaatttc ggctctattg ttttccatct 420
ctcgcaacgg cgggattcct ctatggcgtg tgatgtctgt atctgttact taatccagaa 480
actggcactt gacccaactc tgccacgtgg gtcgttttgc catcgacaga ttgggagatt 540
ttcatagtag aattcagcat gatagctacg taaatgtgtt ccgcaccgtc acaaagtgtt 600
ttctactgtt ctttcttctt tcgttcattc agttgagttg agtgagtgct ttgttcaatg 660
gatcttagct aaatgcatat tttttctctt ggtaaatgaa tgcttgtgat gtcttccaag 720
tgatttcctt tccttcccat atgatgctag gtacctttag tgtcttccta aaaaaaaaaa 780
aaggctcgcc atcaaaacga tattcgttgg cttttttttc tgaattataa atactctttg 840
gtaacttttc atttccaaga acctcttttt tccagttata tcatggtccc ctttcaaagt 900
tattctctac tctttttcat attcattctt tttcatcctt tggtttttta ttcttaactt 960
gtttattatt ctctcttgtt tctatttaca agacaccaat caaaacaaat aaaacatcat 1020
cacaaagctt ggcctgcagg gccagcttac ccttaaattt atttgcacta ctggaaaact 1080
acctgttcca tggccaacac ttgtcactac tttctcttat ggtgttcaat gcttttcccg 1140
ttatccggat catatgaaac ggcatgactt tttcaagagt gccatgcccg aaggttatgt 1200
acaggaacgc actatatctt tcaaagatga cgggaactac aagacgcgtg ctgaagtcaa 1260
gtttgaaggt gatacccttg ttaatcgtat cgagttaaaa ggtattgatt ttaaagaaga 1320
tggaaacatt ctcggacaca aactcgagta caactataac tcacacaatg tatacatcac 1380
ggcagacaaa caaaagaatg gaatcaaagc taacttcaaa attcgccaca acattgaaga 1440
tggatccgtt caactagcag accattatca acaaaatact ccaattggcg atggccctgt 1500
ccttttacca gacaaccatt acctgtcgac acaatctgcc ctttcgaaag atcccaacga 1560
aaagcgtgac cacatggtcc ttcttgagtt tgtaactgct gctgggatta cacatggcat 1620
ggatgagctc tacaaataat gaattcgtcg aggccgttca ggcctcgagg ccgttcaggc 1680
tcgacccggg gatccgcggc ccctaatcag tactgacaat aaaaagattc ttgttttcaa 1740
gaacttgtca tttgtatagt ttttttatat tgtagttgtt ctattttaat caaatgttag 1800
cgtgatttat attttttttc gcctcgacat catctgccca gatgcgaagt taagtgcgca 1860
gaaagtaata tcatgcgtca atcgtatgtg aatgctggtc gctatactgc tgtcgattcg 1920
atactaacgc cgccatccag tgtcgaaaac gagcatgccc atgggttaac tgatcaatgc 1980
atcctgcatg gcgcgcc 1997
<210> 169
<211> 9612
<212> DNA
<213> Artificial Sequence
<220>
<223> Integration vector pEVE2658
<400> 169
tcgcgcgttt cggtgatgac ggtgaaaacc tctgacacat gcagctcccg gagacggtca 60
cagcttgtct gtaagcggat gccgggagca gacaagcccg tcagggcgcg tcagcgggtg 120
ttggcgggtg tcggggctgg cttaactatg cggcatcaga gcagattgta ctgagagtgc 180
accatatgcg gtgtgaaata ccgcacagat gcgtaaggag aaaataccgc atcaggcgcc 240
attcgccatt caggctgcgc aactgttggg aagggcgatc ggtgcgggcc tcttcgctat 300
tacgccagct ggcgaaaggg ggatgtgctg caaggcgatt aagttgggta acgccagggt 360
tttcccagtc acgacgttgt aaaacgacgg ccagtgaatt cgagctccct gcaggctgca 420
acttgtcagc ctttgcgacg atttgtaatg aatccttttg catcgctaga aggagaagat 480
aatatttctt ctgttttttt tttacatatg caacaatttg aaagtcaggt caaagacaga 540
ttccggttcc ccatattcag ataggagaaa aaaacttttg gcaactcatg ttaccaagtc 600
gagaccttaa agttaagtgt cggcctggaa acgcaaaatc ttgtaatctt ttaacgctgc 660
ttttcaaatc acgaacgcaa tcagtacttg tacctaattt tggttttctc gtattgaata 720
gtgaaccata ggtcgaaaat ttaaggccac ataaatccag agcccgcaac ttggataggt 780
atttacttga tttttagttt gctttcaata gtgtcgtgaa attataaagt acgccgcata 840
catatcttga ttagttaaaa atagcagtgt tcaatgatga tttgataggg ttcataactg 900
gtaccagcgt agtacaatta cgattatcca tgaacataaa agtggcttaa atactatata 960
tcagtgaagc ttcaaagtaa gcaaacgaga taccaagatc ttgtaggacc acgatatata 1020
agaagcctag ttccgttgta gcatcacata gaagagagcg acgatgcctc agaaagtcta 1080
cagcaacaga aagctcagtg agcggccgcc agctgaagct tcgtacgctg caggtcgaca 1140
acccttaata taacttcgta taatgtatgc tatacgaagt tattaggtct agagatctgt 1200
ttagcttgcc tcgtccccgc cgggtcaccc ggccagcgac atggaggccc agaataccct 1260
ccttgacagt cttgacgtgc gcagctcagg ggcatgatgt gactgtcgcc cgtacattta 1320
gcccatacat ccccatgtat aatcatttgc atccatacat tttgatggcc gcacggcgcg 1380
aagcaaaaat tacggctcct cgctgcagac ctgcgagcag ggaaacgctc ccctcacaga 1440
cgcgttgaat tgtccccacg ccgcgcccct gtagagaaat ataaaaggtt aggatttgcc 1500
actgaggttc ttctttcata tacttccttt taaaatcttg ctaggataca gttctcacat 1560
cacatccgaa cataaacaac catgggtatg accgaccaag cgacgcccaa cctgccatca 1620
cgagatttcg atcccaccgc cgccttctat gaaaggttgg gcttcggaat cgttttccgg 1680
gacgccggct ggatgatcct ccagcgcggg gatctcatgc tggagttctt cgcccacccc 1740
gggctcgatc ccctcgcgag ttggttcagc tgctgcctga ggctggacga cctcgcggag 1800
ttctaccggc agtgcaaatc cgtcggcatc caggaaacca gcagcggcta tccgcgcatc 1860
catgcccccg aactgcagga gtggggaggc acgatggccg ctttggtcga cccggacggg 1920
acgctcctgc gcctgataca gaacgaattg cttgcaggca tctcatgatc agtactgaca 1980
ataaaaagat tcttgttttc aagaacttgt catttgtata gtttttttat attgtagttg 2040
ttctatttta atcaaatgtt agcgtgattt atattttttt tcgcctcgac atcatctgcc 2100
cagatgcgaa gttaagtgcg cagaaagtaa tatcatgcgt caatcgtatg tgaatgctgg 2160
tcgctatact gctgtcgatt cgatactaac gccgccatcc agtgtcgaaa acgagctctc 2220
gagaaccctt aatataactt cgtataatgt atgctatacg aagttattag gtgatatcag 2280
atccactagt ggcctatgcg gccgcggcgc gcctttgaaa ctaaatttgt atattgtttg 2340
tcagaaatag gacctgatat cctcctttta aacagttgat gagaaccttt ttcgcaagtt 2400
caaggtgctc taatttttaa aatttttact tttcgcgaca caataaagtc ttcacgacgc 2460
taaactatta gtgcacataa tgtagttact tggacgctgt tcaataatgt ataaaattta 2520
tttcctttgc attacgtaca ttatataacc aaatcttaaa aatatagaaa tatgatatgt 2580
gtataataat ataagcaaaa tttacgtatc tttgcttata atatagcttt aatgttcttt 2640
aggtatatat ttaagagcga tttgtccgcg gttacaattc atcgtgaatg gcatcttctt 2700
cgtcagccaa ttcagcgtca gcatcggctt cctcagcagc tttttcctgg gcttcttcgt 2760
acaaggcctt accgtcgacg tcgaagtgac cgttttcctt gatgaagtcg aataaagagt 2820
ccaaggatct tgaaccttgg tacacaacag attcggactt cttaccacct gggtataaga 2880
cgattgttgg gtaaccttca attacgacgc ctctgacatc gttttcagtg tggtctagtt 2940
tagcaatcaa aacgtcggat gtggcgttgg cgtaggtatc agctagttct tggtaagttg 3000
gggccaatct cttacagtga ccacaccatg gggcatagta caaaacaaga acgtccttct 3060
ttgggtcgtt gacgatttcg tcatggttct taccgaccaa ttggaagaca gaggaatctt 3120
ggttctcgaa gatctcttgg gacttcacga ttggggaggc atcacctttc aagaagtcct 3180
taaccaaaga ttcaatagcc ttagactcca acacgatctt gtcgctcaat tcgtcaaacg 3240
cctcttcaga gagttgaggc aaaccgtact tcaagtcttc agtcatgtcg tggatggcaa 3300
atagagggaa ttgttccttc atgttcaagt tgccggcgtg tctgccgaat tttctggcat 3360
cgatgctaac aaagttcatt agacctctgt tctttttggc caactcggta aagagaggct 3420
tgtattcttc caattcttcc tcgtcattgt agaataagta acccaaaggc aaaccgcttt 3480
cgacgtattg ggcgaaaacg gaaccgtcga tttcaccaaa gtagggcaag gcttccactt 3540
gcaaccattt ttcaaaaaca tcagcgtcag cgatatcggc tttcttaccg ttgtatacta 3600
caggctcgtc catggcggag ggcaagtaaa tagaaagctt gaaatcatcg tctgcgtttt 3660
cagcggagac aaagtcgtag tcgttgaagt gtttgttggc catggagtaa aaggtggcgt 3720
tgaagtcggc gtcaatctta ccggattgga cgataactgg agtgacaaaa gtctcgttag 3780
caaggtaagc tggtagatca gcaacaacgg cgacagccgg ttggctttgc ttgatcatga 3840
attggacaat ggcctcggca gttctaggtc cctcgtaatc gatcgagttg ttaacatcgc 3900
tgtttttgaa aatcttcaag cttgggaacc ctggaatgtt gtgttccata cacagatcct 3960
ggttttcagt acagtcgatc tgggccaagg taatgttttt ctcaactaaa gtctcggcgg 4020
ctttaacgta ttcaggagcc atgttcttac agtggccaca ccatggagca aaaaactccg 4080
caagcaccaa gtcgtgcgac tgaatgtact cattgaagga gtcggtggcc aacttaacga 4140
cagcggagtc ttcaggggcc acagcctctt gttgggcgaa aacagaggag gcgagcagca 4200
gggaggacca tgacaggacg gcaccagcag aaaacttcat tttaagcttg tttagttaat 4260
tatagttcgt tgaccgtata ttctaaaaac aagtactcct taaaaaaaaa ccttgaaggg 4320
aataaacaag tagaatagat agagagaaaa atagaaaatg caagagaatt tatatattag 4380
aaagagagaa agaaaaatgg aaaaaaaaaa ataggaaaag ccagaaatag cactagaagg 4440
agcgacacca gaaaagaagg tgatggaacc aatttagcta tatatagtta actaccggct 4500
cgatcatctc tgcctccagc atagtcgaag aagaattttt tttttcttga ggcttctgtc 4560
agcaactcgt attttttctt tcttttttgg tgagcctaaa aagttcccac gttctcttgt 4620
acgacgccgt cacaaacaac cttatgggta atttgtcgcg gtctgggtgt ataaatgtgt 4680
gggtgcaaca tgaatgtacg gaggtagttt gctgattggc ggtctataga taccttggtt 4740
atggcgccct cacagccggc aggggaagcg cctacgcttg acatctacta tatgtaagta 4800
tacggcccac gcgcccagtt cgagtttatc attatcaata ctgccatttc aaagaatacg 4860
taaataatta atagtagtga ttttcctaac tttatttagt caaaaaatta gccttttaat 4920
tctgctgtaa cccgtacatg cccaaaatag ggggcgggtt acacagaata tataacatcg 4980
taggtgtctg ggtgaacagt ttattcctgg catccactaa atataatgga gcccgctttt 5040
taagctggca tccagaaaaa aaaagaatcc cagcaccaaa atattgtttt cttcaccaac 5100
catcagttca taggtccatt ctcttagcgc aactacagag aacaggggca caaacaggca 5160
aaaaacggac acaacctcaa tggagtgatg caacctgcct ggagtaaatg atgacacaag 5220
gcaattgacc cacgcatgta tctatctcat tttcttacac cttctattac cttctgctct 5280
ctctgatttg gaaaaagctg aaaaaaaagg ttgaaaccag ttccctgaaa ttattcccct 5340
acttgactaa taagtatata aagacggtag gtattgattg taattctgta aatctatttc 5400
ttaaacttct taaattctac ttttatagtt agtctttttt ttagttttaa aacaccaaga 5460
acttagtttc gaataaacac acataaacaa acaaaaagct taaaatgcag ttgagcaagg 5520
ctgctgagat gtgttatgag ataacaaact cttacttaca catagaccag aaatctcaga 5580
taatagcaag tacacaagaa gcgatacggt tgacaagaaa atacttacta agtgaaattt 5640
ttgtacgttg gagtccactg aatggggaaa tatcattctc gtacaacgga ggaaaagatt 5700
gccaggtatt actactgtta tatctgagtt gcttatggga atatttcttc attaaggctc 5760
aaaattccca attcgatttc gagtttcaaa gcttccccat gcaaagactt ccaactgttt 5820
tcattgatca agaagaaact ttccctacat tagagaattt tgtactggaa acctcagagc 5880
gatattgcct ttccttatac gaatcacaaa ggcaatctgg tgcatcggtc aatatggcag 5940
acgcatttag agattttata aagatatacc ctgagaccga agctatagtg ataggtatta 6000
gacacacaga cccatttggt gaagcattaa agcctattca aagaacagat tctaactggc 6060
ctgattttat gaggttgcaa cctctcttac actgggactt aaccaatata tggagtttct 6120
tactgtattc taatgagcca atttgtggac tatatggtaa aggtttcaca tcaatcggcg 6180
gaattaacaa ctcattgcct aacccacact tgagaaagga ctccaataat ccagccttgc 6240
attttgaatg ggaaatcatt catgcatttg gcaaggacgc agaaggcgaa cgtagttccg 6300
ctataaacac gtcacctatt tccgtggtgg ataaggaaag attcagcaaa taccatgaca 6360
attactatcc tggctggtat ttggttgatg acactttaga gagagcaggc aggatcaaga 6420
attaaccgcg gcacgtccga cggcggcccg acgggtccga ggcctcggag atccgtcccc 6480
cttttccttt gtcgatatca tgtaattagt tatgtcacgc ttacattcac gccctccccc 6540
cacatccgct ctaaccgaaa aggaaggagt tagacaacct gaagtctagg tccctattta 6600
tttttttata gttatgttag tattaagaac gttatttata tttcaaattt ttcttttttt 6660
tctgtacaga cgcgccgcga gggcacatac atacagtggg acctcatgag atccagaaaa 6720
aaagcaaagg aaagttcgat cgaaacatta gctcttagat attacagacg tgaggtatct 6780
cttccttgaa gagaatctgg ccctgatcaa aaactttatt ttgatactgt tcgaaaagag 6840
aaataaggac aacgttctaa tttatacaaa tataaccagt aggctttatc gtaaaatcct 6900
cgaacttgct tctcatcaca gaagtataaa aagtaaagag gtcaagcaag aaacgaataa 6960
ccaccaaatt tttttcaatt gaaaagcgaa acgtaatgga aattcacctg ctttgaatcg 7020
ttgttgaatt cttatcatca gtcattattt tcctctttta aagcttaagt caaccgcatc 7080
taagtttaac acttcgttcc cgttttgaag tttatcaaaa ttccaaaata cagtagttag 7140
gtgctcgtga caaacctttt tccaaagtgt cattagactt catggaacta ctacacttct 7200
ggggaacgaa aagctaccat ttgctcatca ttggataatt tctttttttt tttttgatgc 7260
atccaaactt ggaccccttt tgaatgtcga gtatgaagta tcaattttag ggcaagtagt 7320
tttacaataa ttttggtgca cttttacgcc tctgctgacc tgcagggcat gcaagcttgg 7380
cgtaatcatg gtcatagctg tttcctgtgt gaaattgtta tccgctcaca attccacaca 7440
acatacgagc cggaagcata aagtgtaaag cctggggtgc ctaatgagtg agctaactca 7500
cattaattgc gttgcgctca ctgcccgctt tccagtcggg aaacctgtcg tgccagctgc 7560
attaatgaat cggccaacgc gcggggagag gcggtttgcg tattgggcgc tcttccgctt 7620
cctcgctcac tgactcgctg cgctcggtcg ttcggctgcg gcgagcggta tcagctcact 7680
caaaggcggt aatacggtta tccacagaat caggggataa cgcaggaaag aacatgtgag 7740
caaaaggcca gcaaaaggcc aggaaccgta aaaaggccgc gttgctggcg tttttccata 7800
ggctccgccc ccctgacgag catcacaaaa atcgacgctc aagtcagagg tggcgaaacc 7860
cgacaggact ataaagatac caggcgtttc cccctggaag ctccctcgtg cgctctcctg 7920
ttccgaccct gccgcttacc ggatacctgt ccgcctttct cccttcggga agcgtggcgc 7980
tttctcatag ctcacgctgt aggtatctca gttcggtgta ggtcgttcgc tccaagctgg 8040
gctgtgtgca cgaacccccc gttcagcccg accgctgcgc cttatccggt aactatcgtc 8100
ttgagtccaa cccggtaaga cacgacttat cgccactggc agcagccact ggtaacagga 8160
ttagcagagc gaggtatgta ggcggtgcta cagagttctt gaagtggtgg cctaactacg 8220
gctacactag aaggacagta tttggtatct gcgctctgct gaagccagtt accttcggaa 8280
aaagagttgg tagctcttga tccggcaaac aaaccaccgc tggtagcggt ggtttttttg 8340
tttgcaagca gcagattacg cgcagaaaaa aaggatctca agaagatcct ttgatctttt 8400
ctacggggtc tgacgctcag tggaacgaaa actcacgtta agggattttg gtcatgagat 8460
tatcaaaaag gatcttcacc tagatccttt taaattaaaa atgaagtttt aaatcaatct 8520
aaagtatata tgagtaaact tggtctgaca gttaccaatg cttaatcagt gaggcaccta 8580
tctcagcgat ctgtctattt cgttcatcca tagttgcctg actccccgtc gtgtagataa 8640
ctacgatacg ggagggctta ccatctggcc ccagtgctgc aatgataccg cgagacccac 8700
gctcaccggc tccagattta tcagcaataa accagccagc cggaagggcc gagcgcagaa 8760
gtggtcctgc aactttatcc gcctccatcc agtctattaa ttgttgccgg gaagctagag 8820
taagtagttc gccagttaat agtttgcgca acgttgttgc cattgctaca ggcatcgtgg 8880
tgtcacgctc gtcgtttggt atggcttcat tcagctccgg ttcccaacga tcaaggcgag 8940
ttacatgatc ccccatgttg tgcaaaaaag cggttagctc cttcggtcct ccgatcgttg 9000
tcagaagtaa gttggccgca gtgttatcac tcatggttat ggcagcactg cataattctc 9060
ttactgtcat gccatccgta agatgctttt ctgtgactgg tgagtactca accaagtcat 9120
tctgagaata gtgtatgcgg cgaccgagtt gctcttgccc ggcgtcaata cgggataata 9180
ccgcgccaca tagcagaact ttaaaagtgc tcatcattgg aaaacgttct tcggggcgaa 9240
aactctcaag gatcttaccg ctgttgagat ccagttcgat gtaacccact cgtgcaccca 9300
actgatcttc agcatctttt actttcacca gcgtttctgg gtgagcaaaa acaggaaggc 9360
aaaatgccgc aaaaaaggga ataagggcga cacggaaatg ttgaatactc atactcttcc 9420
tttttcaata ttattgaagc atttatcagg gttattgtct catgagcgga tacatatttg 9480
aatgtattta gaaaaataaa caaatagggg ttccgcgcac atttccccga aaagtgccac 9540
ctgacgtcta agaaaccatt attatcatga cattaaccta taaaaatagg cgtatcacga 9600
ggccctttcg tc 9612
<210> 170
<211> 10201
<212> DNA
<213> Artificial Sequence
<220>
<223> Integration vector pEVE2682
<400> 170
tcgcgcgttt cggtgatgac ggtgaaaacc tctgacacat gcagctcccg gagacggtca 60
cagcttgtct gtaagcggat gccgggagca gacaagcccg tcagggcgcg tcagcgggtg 120
ttggcgggtg tcggggctgg cttaactatg cggcatcaga gcagattgta ctgagagtgc 180
accatatgcg gtgtgaaata ccgcacagat gcgtaaggag aaaataccgc atcaggcgcc 240
attcgccatt caggctgcgc aactgttggg aagggcgatc ggtgcgggcc tcttcgctat 300
tacgccagct ggcgaaaggg ggatgtgctg caaggcgatt aagttgggta acgccagggt 360
tttcccagtc acgacgttgt aaaacgacgg ccagtgaatt cgagctccct gcaggtcaat 420
caaagcaacc cacaaatcct aggctgaatc atgatatcga tggaagcaat caacaatttt 480
atcaagaccg caccaaagca cgactatctg acaggcggag ttcatcattc tggtaatgta 540
gacgtgttac aattaagcgg caataaagaa gatggtagtt tagtatggaa ccatactttt 600
gttgatgtag acaacaatgt ggtagctaag tttgaagacg ctctcgaaaa acttgaaagt 660
ttgcaccggc gctcatcctc atccacaggc aatgaagaac acgctaacgt ttaaccgagg 720
ggagtcactt cataatgatg tgagaaataa gtgaatattg taataattgt tgggactcca 780
ttgtcaacaa aagctataat gtaggtatac agtatatact agaagttctc ctcgaggatc 840
ttggaatcca caaaagggag tcgataaatc tatataataa aaattacttt atcttctttc 900
gttttatacg ttgtcgttta ttatcctatt acgttatcaa tcttcgcatt tcagctttca 960
ttagatttga tgactgtttc tcaaacttta tgtcattttc ttacccgcag cggccgccag 1020
ctgaagcttc gtacgctgca ggtcgacaac ccttaatata acttcgtata atgtatgcta 1080
tacgaagtta ttaggtctag agatctgttt agcttgcctt gtccccgccg ggtcacccgg 1140
ccagcgacat ggaggcccag aataccctcc ttgacagtct tgacgtgcgc agctcagggg 1200
catgatgtga ctgtcgcccg tacatttagc ccatacatcc ccatgtataa tcatttgcat 1260
ccatacattt tgatggccgc acggcgcgaa gcaaaaatta cggctcctcg ctgcagacct 1320
gcgagcaggg aaacgctccc ctcacagacg cgttgaattg tccccacgcc gcgcccctgt 1380
agagaaatat aaaaggttag gatttgccac tgaggttctt ctttcatata cttcctttta 1440
aaatcttgct aggatacagt tctcacatca catccgaaca taaacaacca tgggtaaaaa 1500
gcctgaactc accgcgacgt ctgtcgagaa gtttctgatc gaaaagttcg acagcgtctc 1560
cgacctgatg cagctctcgg agggcgaaga atctcgtgct ttcagcttcg atgtaggagg 1620
gcgtggatat gtcctgcggg taaatagctg cgccgatggt ttctacaaag atcgttatgt 1680
ttatcggcac tttgcatcgg ccgcgctccc gattccggaa gtgcttgaca ttggggaatt 1740
cagcgagagc ctgacctatt gcatctcccg ccgtgcacag ggtgtcacgt tgcaagacct 1800
gcctgaaacc gaactgcccg ctgttctgca gccggtcgcg gaggccatgg atgcgatcgc 1860
tgcggccgat cttagccaga cgagcgggtt cggcccattc ggaccgcaag gaatcggtca 1920
atacactaca tggcgtgatt tcatatgcgc gattgctgat ccccatgtgt atcactggca 1980
aactgtgatg gacgacaccg tcagtgcgtc cgtcgcgcag gctctcgatg agctgatgct 2040
ttgggccgag gactgccccg aagtccggca cctcgtgcac gcggatttcg gctccaacaa 2100
tgtcctgacg gacaatggcc gcataacagc ggtcattgac tggagcgagg cgatgttcgg 2160
ggattcccaa tacgaggtcg ccaacatctt cttctggagg ccgtggttgg cttgtatgga 2220
gcagcagacg cgctacttcg agcggaggca tccggagctt gcaggatcgc cgcggctccg 2280
ggcgtatatg ctccgcattg gtcttgacca actctatcag agcttggttg acggcaattt 2340
cgatgatgca gcttgggcgc agggtcgatg cgacgcaatc gtccgatccg gagccgggac 2400
tgtcgggcgt acacaaatcg cccgcagaag cgcggccgtc tggaccgatg gctgtgtaga 2460
agtactcgcc gatagtggaa accgacgccc cagcactcgt ccgagggcaa aggaataatc 2520
agtactgaca ataaaaagat tcttgttttc aagaacttgt catttgtata gtttttttat 2580
attgtagttg ttctatttta atcaaatgtt agcgtgattt atattttttt tcgcctcgac 2640
atcatctgcc cagatgcgaa gttaagtgcg cagaaagtaa tatcatgcgt caatcgtatg 2700
tgaatgctgg tcgctatact gctgtcgatt cgatactaac gccgccatcc agtgtcgaaa 2760
acgagctctc gagaaccctt aatataactt cgtataatgt atgctatacg aagttattag 2820
gtgatatcag atccactagt ggcctatgcg gccgcggcgc gccgaaggaa aatgagaaat 2880
atcgagggag acgattcaga ggagcaggac aaactataac cgactgtttg ttggaggatg 2940
ccgtacataa cgaacactgc tgaagctacc atgtctacag tttagaggaa tgggtacaac 3000
tcacaggcga gggatggtgt tcactcgtgc tagcaaacgc ggtgggagca aaaagtagaa 3060
tattatcttt tattcgtgaa acttcgaaca ctgtcatcta aagatgctat atactaatat 3120
aggcatactt gataatgaaa actataaatc gtaaagacat aagagatccg cggctagtac 3180
tgggtgatga cgccaggctc tgatccatct ggggttagtg gtagttttgt ctccctaacg 3240
gtggttccgg cggtcttggt cagcttggac agctcttgga cggtggctga aaggccaggg 3300
tccacatgga cagagttgaa ggatgaggta acccaatcac cagtttccaa agattgcaag 3360
tagacggaca agtatggctt gttctttctg taagagaatg agatgtaagc gtgcaagtag 3420
ttcaaagttt cgtgatcagc atgtgggtaa tgagagcaca attcagcttt gtactgtcta 3480
gccatttcgt gccaaccacg acgttcgaag aaagtagcca aagcatcagc aatttgcatg 3540
tcgtttctac cgaaagtagt aaagtaaact tgtggttctg gaactggatc atcatgatgg 3600
atggtgtagt tggccatcaa tggcaattgt tcatctggaa tagtaccgat tggcaaatat 3660
ggagctggat aagattgtaa accagctggc aatctgatca attcccacaa ttctctaatc 3720
atttccaaac cagccaaagt agaagcatca gttctttcac cacccatagt ccacaaatct 3780
tccaaggctt ccaaagtgac cattctttcc aacaagtaga ttttgattct ggacttggta 3840
gggtcaatca aatcgcaaga caataatctt ggagaggcag tagaagatgg acctctagaa 3900
taaacgtatt gttccaaggt atccaatggc tttctcaatt ctgggtattg cttagaccat 3960
ctgaaagcag aaccaaaaac caattcgtgg atacttctac cagtagccaa agatttcaag 4020
gctgggtaga tgtaagtctt aacaacgaat tcaccatctt gcaagtccaa agccaatttg 4080
ttttgggttc tgatttgttc accgaccaag ttgttcttgt acaagtattc ggattcttca 4140
gcagacaaag tcaagtcgtt cttgaagtgt ctgaacaaat ccaaatcgat accggattgt 4200
aaagccatca attgttccaa tgagtcccaa acagcatgag tattgaatgg atctctagca 4260
gtaccagtag cagcgttaat aggttcgtaa gtgtatctaa ccaatggacc agaacagttc 4320
aaagacaatt cgaatggagt accatatctg gtcaaaatgg acaaccatct tggcttatca 4380
ttagttgggt aaacacccaa gaatggaatg acgttcttct tgtagatcaa caagtgcttg 4440
tactgagcgt gtaagttgta gttggtagat tgcaacattt gagcaaacat tggagcagta 4500
gaatgccacc acaattcttc atcttgattg gcgaaatcca aagcaacaga caaagttctg 4560
tagacttcgc aatcagaagc attaccagtc ttaatacctt gaccagcagt cattttaagc 4620
tttgttttat atttgttgta aaaagtagat aattacttcc ttgatgatct gtaaaaaaga 4680
gaaaaagaaa gcatctaaga acttgaaaaa ctacgaatta gaaaagacca aatatgtatt 4740
tcttgcattg accaatttat gcaagtttat atatatgtaa atgtaagttt cacgaggttc 4800
tactaaacta aaccaccccc ttggttagaa gaaaagagtg tgtgagaaca ggctgttgtt 4860
gtcacacgat tcggacaatt ctgtttgaaa gagagagagt aacagtacga tcgaacgaac 4920
tttgctctgg agatcacagt gggcatcata gcatgtggta ctaaaccctt tcccgccatt 4980
ccagaacctt cgattgcttg ttacaaaacc tgtgagccgt cgctaggacc ttgttgtgtg 5040
acgaaattgg aagctgcaat caataggaag acaggaagtc gagcgtgtct gggttttttc 5100
agttttgttc tttttgcaaa caaatcacga gcgacggtaa tttctttctc gataagaggc 5160
cacgtgcttt atgagggtaa catcaattca agaaggaggg aaacacttcc tttttctggc 5220
cctgataata gtatgagggt gaagccaaaa taaaggattc gcgcccaaat cggcatcttt 5280
aaatgcaggt atgcgatagt tcctcactct ttccttactc acgagtaatt cttgcaaatg 5340
cctattatgc agatgttata atatctgtgc gtacgcgccc agttcgagtt tatcattatc 5400
aatactgcca tttcaaagaa tacgtaaata attaatagta gtgattttcc taactttatt 5460
tagtcaaaaa attagccttt taattctgct gtaacccgta catgcccaaa atagggggcg 5520
ggttacacag aatatataac atcgtaggtg tctgggtgaa cagtttattc ctggcatcca 5580
ctaaatataa tggagcccgc tttttaagct ggcatccaga aaaaaaaaga atcccagcac 5640
caaaatattg ttttcttcac caaccatcag ttcataggtc cattctctta gcgcaactac 5700
agagaacagg ggcacaaaca ggcaaaaaac ggacacaacc tcaatggagt gatgcaacct 5760
gcctggagta aatgatgaca caaggcaatt gacccacgca tgtatctatc tcattttctt 5820
acaccttcta ttaccttctg ctctctctga tttggaaaaa gctgaaaaaa aaggttgaaa 5880
ccagttccct gaaattattc ccctacttga ctaataagta tataaagacg gtaggtattg 5940
attgtaattc tgtaaatcta tttcttaaac ttcttaaatt ctacttttat agttagtctt 6000
ttttttagtt ttaaaacacc aagaacttag tttcgaataa acacacataa acaaacaaaa 6060
agcttaaaat gaccattacc gaaacttcta agccaacctt gagaagaatt ccaagatcag 6120
ctggtgatga agctattttc caagtcttgc aagaagatgg cgttgttgtt atcgaaggtt 6180
tcatgtctgc tgaccaagtt agaagattca acggtgaaat tgatccacac atgaagcaat 6240
gggaattggg tcaaaaatcc taccaagaat cttacttggc tggtatgaga caattgtcat 6300
ctttgccttt gttctccaag ttgttcagag atgaattgat gaacgacgaa ttattgcacg 6360
gtttgtgcaa gagattattc ggtccagaat ctggtgatta ctggttgact acttcttctg 6420
ttttggaaac tgaaccaggt taccatggtc aagaattgca tagagaacat gatggtattc 6480
caatctgtac taccttgggt agacaatctc cagaatctat gttgaacttc ttgactgctt 6540
tgactgattt cactgctgaa aatggtgcta ctagagtttt gccaggttct catttgtggg 6600
aagatttttc tgctccacca ccaaaagctg atactgctat tccagctgtt atgaatccag 6660
gtgatgctgt tttgtttact ggtaaaactt tacatggtgc cggtaagaac aacactactg 6720
attttttgag aagaggtttc ccattgatca tgcaatcttg tcaattcact ccagttgaag 6780
ctagtgttgc tttgccaaga gaattggttg aaactatgac tccattggcc caaaaaatgg 6840
ttggttggag aactgtttct gctaagggtg ttgatatttg gacctacgat ttgaaggatt 6900
tggctaccgg tattgacttg aagtctaatc aagttgctaa gaaggcctaa ccgcggcacg 6960
tccgacggcg gcccgacggg tccgaggcct cggagatccg tccccctttt cctttgtcga 7020
tatcatgtaa ttagttatgt cacgcttaca ttcacgccct ccccccacat ccgctctaac 7080
cgaaaaggaa ggagttagac aacctgaagt ctaggtccct atttattttt ttatagttat 7140
gttagtatta agaacgttat ttatatttca aatttttctt ttttttctgt acagacgcgc 7200
ccatggaaaa tgcaaccgat aaaccattat aaatcttcgc ggttatctgg cattgttatt 7260
aaccaaaaaa atgccggcct attacaagct actgttcaat aaatattgtt gtaatgaaga 7320
cggtccaact gtacaaatac agcaaactgt catatataag gagtcttatg tgacagcact 7380
tgcgttattg tcagccggag tatgtctttg tcgcattctg ggctttttac tttctgctca 7440
gaaggaagta cgaacaagaa aaaaaaatca ccaatgcttc ccttttcagt attagtttca 7500
tatttgttta cgttcaaact cgtcgtttgc gcgataacct ctaaaaaagt caattacgta 7560
actatatcaa tcagagaatg caaaaagcac tatcataaaa atgtgtctag gggatgtgag 7620
acatgtcaat tataagaagt gatggtgtca tagtatatat atcataaaag attatcaaag 7680
tttcaatcct ttgtattttc tagtttagcg ccaacttttg acaaaaccta aactttagat 7740
aatcatcatt cttacaattt ttatctggat ggcaataatc tcctatataa agcccagata 7800
aactgtaaaa agaatccatc actatttgaa aaaaagtcat ctggcacgtt taattatcag 7860
agcagaaatg atgaagggtg ttagcgccgt ccactgatgt gcctggtagt catgatttac 7920
gtataactaa cacatcatga ggacggccct gcagggcatg caagcttggc gtaatcatgg 7980
tcatagctgt ttcctgtgtg aaattgttat ccgctcacaa ttccacacaa catacgagcc 8040
ggaagcataa agtgtaaagc ctggggtgcc taatgagtga gctaactcac attaattgcg 8100
ttgcgctcac tgcccgcttt ccagtcggga aacctgtcgt gccagctgca ttaatgaatc 8160
ggccaacgcg cggggagagg cggtttgcgt attgggcgct cttccgcttc ctcgctcact 8220
gactcgctgc gctcggtcgt tcggctgcgg cgagcggtat cagctcactc aaaggcggta 8280
atacggttat ccacagaatc aggggataac gcaggaaaga acatgtgagc aaaaggccag 8340
caaaaggcca ggaaccgtaa aaaggccgcg ttgctggcgt ttttccatag gctccgcccc 8400
cctgacgagc atcacaaaaa tcgacgctca agtcagaggt ggcgaaaccc gacaggacta 8460
taaagatacc aggcgtttcc ccctggaagc tccctcgtgc gctctcctgt tccgaccctg 8520
ccgcttaccg gatacctgtc cgcctttctc ccttcgggaa gcgtggcgct ttctcatagc 8580
tcacgctgta ggtatctcag ttcggtgtag gtcgttcgct ccaagctggg ctgtgtgcac 8640
gaaccccccg ttcagcccga ccgctgcgcc ttatccggta actatcgtct tgagtccaac 8700
ccggtaagac acgacttatc gccactggca gcagccactg gtaacaggat tagcagagcg 8760
aggtatgtag gcggtgctac agagttcttg aagtggtggc ctaactacgg ctacactaga 8820
aggacagtat ttggtatctg cgctctgctg aagccagtta ccttcggaaa aagagttggt 8880
agctcttgat ccggcaaaca aaccaccgct ggtagcggtg gtttttttgt ttgcaagcag 8940
cagattacgc gcagaaaaaa aggatctcaa gaagatcctt tgatcttttc tacggggtct 9000
gacgctcagt ggaacgaaaa ctcacgttaa gggattttgg tcatgagatt atcaaaaagg 9060
atcttcacct agatcctttt aaattaaaaa tgaagtttta aatcaatcta aagtatatat 9120
gagtaaactt ggtctgacag ttaccaatgc ttaatcagtg aggcacctat ctcagcgatc 9180
tgtctatttc gttcatccat agttgcctga ctccccgtcg tgtagataac tacgatacgg 9240
gagggcttac catctggccc cagtgctgca atgataccgc gagacccacg ctcaccggct 9300
ccagatttat cagcaataaa ccagccagcc ggaagggccg agcgcagaag tggtcctgca 9360
actttatccg cctccatcca gtctattaat tgttgccggg aagctagagt aagtagttcg 9420
ccagttaata gtttgcgcaa cgttgttgcc attgctacag gcatcgtggt gtcacgctcg 9480
tcgtttggta tggcttcatt cagctccggt tcccaacgat caaggcgagt tacatgatcc 9540
cccatgttgt gcaaaaaagc ggttagctcc ttcggtcctc cgatcgttgt cagaagtaag 9600
ttggccgcag tgttatcact catggttatg gcagcactgc ataattctct tactgtcatg 9660
ccatccgtaa gatgcttttc tgtgactggt gagtactcaa ccaagtcatt ctgagaatag 9720
tgtatgcggc gaccgagttg ctcttgcccg gcgtcaatac gggataatac cgcgccacat 9780
agcagaactt taaaagtgct catcattgga aaacgttctt cggggcgaaa actctcaagg 9840
atcttaccgc tgttgagatc cagttcgatg taacccactc gtgcacccaa ctgatcttca 9900
gcatctttta ctttcaccag cgtttctggg tgagcaaaaa caggaaggca aaatgccgca 9960
aaaaagggaa taagggcgac acggaaatgt tgaatactca tactcttcct ttttcaatat 10020
tattgaagca tttatcaggg ttattgtctc atgagcggat acatatttga atgtatttag 10080
aaaaataaac aaataggggt tccgcgcaca tttccccgaa aagtgccacc tgacgtctaa 10140
gaaaccatta ttatcatgac attaacctat aaaaataggc gtatcacgag gccctttcgt 10200
c 10201
<210> 171
<211> 12651
<212> DNA
<213> Artificial Sequence
<220>
<223> Integration vector pEVE2723
<400> 171
tcgcgcgttt cggtgatgac ggtgaaaacc tctgacacat gcagctcccg gagacggtca 60
cagcttgtct gtaagcggat gccgggagca gacaagcccg tcagggcgcg tcagcgggtg 120
ttggcgggtg tcggggctgg cttaactatg cggcatcaga gcagattgta ctgagagtgc 180
accatatgcg gtgtgaaata ccgcacagat gcgtaaggag aaaataccgc atcaggcgcc 240
attcgccatt caggctgcgc aactgttggg aagggcgatc ggtgcgggcc tcttcgctat 300
tacgccagct ggcgaaaggg ggatgtgctg caaggcgatt aagttgggta acgccagggt 360
tttcccagtc acgacgttgt aaaacgacgg ccagtgaatt cgagctccct gcaggttcgt 420
gaaacacgtg ggataccggt taggtaatat tacagtttcc ttacacactt actaaatacg 480
caacagagca tgttattaat cgtatattta agtaataata acgtattgac cattcctaag 540
actgaagcag taccaagaaa tggcactttt tgtttatatg acatagtaaa agtgttcatt 600
tggcagaaaa caagggtatc tggatactgc aatcctcatt agttgcggag agctttttta 660
aagttttatt acttcgataa tgctttcctc cagaactggg cgcgcactga agccataaac 720
tgagtggttg tccctgtgta tattctacaa tcgcctattt gaaacttctg ggtgataata 780
tcgatttagc gattgggttt ctcccttctg agattctgga taaatgagat gctaccctca 840
cagtaaaaac agtacaaaac ctttgtatag ttctagtcaa atatgaactc cttcattcat 900
ggattgtata ctattgttct tgttgaaaga ttcctcttgt gattttcatg gtgattttaa 960
gcctttttta ttgaaagaat taaatattca ctaggctgcg atacgataga caaacgaagt 1020
gattgaaacc cgaattaacg gagcggccgc cagctgaagc ttcgtacgct gcaggtcgac 1080
aacccttaat ataacttcgt ataatgtatg ctatacgaag ttattaggtc tagagatctg 1140
tttagcttgc cttgtccccg ccgggtcacc cggccagcga catggaggcc cagaataccc 1200
tccttgacag tcttgacgtg cgcagctcag gggcatgatg tgactgtcgc ccgtacattt 1260
agcccataca tccccatgta taatcatttg catccataca ttttgatggc cgcacggcgc 1320
gaagcaaaaa ttacggctcc tcgctgcaga cctgcgagca gggaaacgct cccctcacag 1380
acgcgttgaa ttgtccccac gccgcgcccc tgtagagaaa tataaaaggt taggatttgc 1440
cactgaggtt cttctttcat atacttcctt ttaaaatctt gctaggatac agttctcaca 1500
tcacatccga acataaacaa ccatgggtac cactcttgac gacacggctt accggtaccg 1560
caccagtgtc ccgggggacg ccgaggccat cgaggcactg gatgggtcct tcaccaccga 1620
caccgtcttc cgcgtcaccg ccaccgggga cggcttcacc ctgcgggagg tgccggtgga 1680
cccgcccctg accaaggtgt tccccgacga cgaatcggac gacgaatcgg acgacgggga 1740
ggacggcgac ccggactccc ggacgttcgt cgcgtacggg gacgacggcg acctggcggg 1800
cttcgtggtc gtctcgtact ccggctggaa ccgccggctg accgtcgagg acatcgaggt 1860
cgccccggag caccgggggc acggggtcgg gcgcgcgttg atggggctcg cgacggagtt 1920
cgcccgcgag cggggcgccg ggcacctctg gctggaggtc accaacgtca acgcaccggc 1980
gatccacgcg taccggcgga tggggttcac cctctgcggc ctggacaccg ccctgtacga 2040
cggcaccgcc tcggacggcg agcaggcgct ctacatgagc atgccctgcc cctaatcagt 2100
actgacaata aaaagattct tgttttcaag aacttgtcat ttgtatagtt tttttatatt 2160
gtagttgttc tattttaatc aaatgttagc gtgatttata ttttttttcg cctcgacatc 2220
atctgcccag atgcgaagtt aagtgcgcag aaagtaatat catgcgtcaa tcgtatgtga 2280
atgctggtcg ctatactgct gtcgattcga tactaacgcc gccatccagt gtcgaaaacg 2340
agctctcgag aacccttaat ataacttcgt ataatgtatg ctatacgaag ttattaggtg 2400
atatcagatc cactagtggc ctatgcggcc gcggcgcgcc atgcagggat atcagatctg 2460
ggccgtatac ttacatatag tagatgtcaa gcgtaggcgc ttcccctgcc ggctgtgagg 2520
gcgccataac caaggtatct atagaccgcc aatcagcaaa ctacctccgt acattcatgt 2580
tgcacccaca catttataca cccagaccgc gacaaattac ccataaggtt gtttgtgacg 2640
gcgtcgtaca agagaacgtg ggaacttttt aggctcacca aaaaagaaag aaaaaatacg 2700
agttgctgac agaagcctca agaaaaaaaa aattcttctt cgactatgct ggaggcagag 2760
atgatcgagc cggtagttaa ctatatatag ctaaattggt tccatcacct tcttttctgg 2820
tgtcgctcct tctagtgcta tttctggctt ttcctatttt tttttttcca tttttctttc 2880
tctctttcta atatataaat tctcttgcat tttctatttt tctctctatc tattctactt 2940
gtttattccc ttcaaggttt ttttttaagg agtacttgtt tttagaatat acggtcaacg 3000
aactataatt aactaaacaa gcttaaaatg ggtcaatcca gaggtatttt gggtggtgtt 3060
agacaattga tcttggttat tttggttggt gcctacttgt ctagattgtc tgctgttgat 3120
gatgatagac atgattgcag atgtagacca ggtgaaccat gttggccaac tgtttctcat 3180
tggtctactt tgaacgaatc tattggtggt gctttggctc atgttaagcc aattgctcat 3240
gtttgtcatc aatccggtca agattcttct gcttgcgaac aagttttaca agaatccttg 3300
gattctaagt ggcgtgcttc tcatactggt gctttacaag attgggtttg ggaaggtggt 3360
gctgaatcta atcaaacttg ttactacttg agatcaggtc cagctggttc ttgtcatcaa 3420
ggtagaattc cattatattc cgctgctgtt aagtccgctt ctgatgttca aaaggttgtt 3480
gatttcacca gacaacacaa cttgagattg gttattagaa acaccggtca tgatggttct 3540
ggtagatcat ctggtccaga ttctgttgaa attcataccc atcacttgaa ctccgttcaa 3600
taccatccaa attttagacc agctggttcc tctgaaagac aatctgctcc aggtcaacca 3660
gctgttactg ttggtgctgg tattttgttg ggtgacttgt atgctagagg tgcttctgaa 3720
ggttggatag ttgttggtgg tgaatgtcca acagttggtg ctgctggtgg ttttttacaa 3780
ggtggtggtg tttcttcttg gttgtcttat gctcatggtt tggctgttga taacgtcttg 3840
gaatacgaag ttgttactgc taagggtgaa atcgttattg ctaacgctca tcaaaaccca 3900
gatttgtttt gggctttgag aggcggtggt ggtggtactt ttggtgttgt tactcaagct 3960
acattgcaag ttcatccaga tttgccagtt tctgttgctg atgctgttgt tactggttct 4020
agagctgatg ctactttttg gtcacatggt gttgctgctt tgttgagagc tttacaattc 4080
ttgaacaacc atggtactgc cggtcaattc attttgagaa acactgatga tgataccgtc 4140
caagctagtt tgactatgta cttctctaac ttgactgttc cagctgttgc tgacgaaaga 4200
atggatcctt tgagaagagc tttggaacat aatggtcatc cataccaatt gacctctaga 4260
ttcttgccac aaatctcttc tactttcaga cataccgctg atagataccc agaagattac 4320
ggtattttga tgggttccgt tttggtttcc ttggacttgt ttaattctgc tactggtcca 4380
gctgctttgg cacaacattt tgctagattg ccaatgactc cagatgattt gttgttcact 4440
tctaacttgg gtggtagagt ctcttcattg aatagagatc cagcttctac tgctatgcat 4500
ccaggttgga gagatgctgc tcaattattg aatttcgtta gaggtgttgg tgctccatct 4560
ttggctgcta aagctactgc tttacatgaa ttgcaaaccg ttcaaatggc cagattatac 4620
gaaattgaac cagccttcca aatctcctac agaaatttgg gtgatccatc cgaaagaaga 4680
tccagagaag tttattgggg ttctaactac gctagattgg ttgaagttaa gagaagatgg 4740
gatccagaag gtttgttctt ctccaaattg ggtattgatg gtgatgcttg ggatgctgaa 4800
ggtatgtgta gacaaacaag acaaggtgct tggaatgttg ctgttaagtg ggttcaatct 4860
ttcttgggtt ctttgtctgc ttccgttgtt tgaccgcgga caaatcgctc ttaaatatat 4920
acctaaagaa cattaaagct atattataag caaagatacg taaattttgc ttatattatt 4980
atacacatat catatttcta tatttttaag atttggttat ataatgtacg taatgcaaag 5040
gaaataaatt ttatacatta ttgaacagcg tccaagtaac tacattatgt gcactaatag 5100
tttagcgtcg tgaagacttt attgtgtcgc gaaaagtaaa aattttaaaa attagagcac 5160
cttgaacttg cgaaaaaggt tctcatcaac tgtttaaaag gaggatatca ggtcctattt 5220
ctgacaaaca atatacaaat ttagtttcaa agcatgccca tgggttaact gatcaatgca 5280
tcctgcatgg cgcgccgaag gaaaatgaga aatatcgagg gagacgattc agaggagcag 5340
gacaaactat aaccgactgt ttgttggagg atgccgtaca taacgaacac tgctgaagct 5400
accatgtcta cagtttagag gaatgggtac aactcacagg cgagggatgg tgttcactcg 5460
tgctagcaaa cgcggtggga gcaaaaagta gaatattatc ttttattcgt gaaacttcga 5520
acactgtcat ctaaagatgc tatatactaa tataggcata cttgataatg aaaactataa 5580
atcgtaaaga cataagagat ccgcggtcac aatctagctg gaataaaaga ttcttcagct 5640
ggagcttcag acttttgcaa caaggtttca acaccttttt gaattctagc agccaaagct 5700
ggagaaacca acttccaggt ttcgtaaact tgcttttgaa tttcggctct ggtttgagca 5760
acagattcaa ccatgttttg gaccatggta tcttggaatt cttgacctct aaaatctggc 5820
aaaacttcgt agaaatctct ggcgaacttg taatcatgtt cagtagtttg caaccaggac 5880
ttggattgga tttctttcaa ccattcatta tactttggat ctggttctct aggttcggca 5940
aatctaactg gttgttgttg agtcaagaaa cctggatgag caccataatt agcatcaaaa 6000
gtaccttgac catctctagc aattgggtta aaggtatgag cggatctatt aactgggatt 6060
tgttgatggt tagcacccaa tctatatctt tgagcgtctg ggtaagcgaa aattctagct 6120
tgtaaaattg gatcttcaga tggagcaaca cctggaacca tgttagatgg agagtaagcc 6180
aattgttcaa tttcggcgaa gtagttagct gggtttcttt tcaaagtcaa cttaccgaaa 6240
acctttggtg ggatcaaagt aacatctttt gggtaagtac ccaagttcca gtgcttagta 6300
acatccaaaa tgttgtaacc caagtttgga gcatcttttg gatcaacaac ttgaacagaa 6360
gcagtccaag ttgggtattc acctctttca atagcttcat acaagtctct agtagcgtga 6420
tctggatcca aatcatttgt acctttagct tgttgaccat cggtgaaatt tggacctcta 6480
tcagatgaca agaagatgtg gacgtactta aaagaaccat ctggcttaac ccatttgtaa 6540
gcatgagcca tataaccaga catggatctc caattgaaca tagtaccgaa atcagaaaat 6600
tgcaacaaga ccatgtgcaa ggattcatga ttgttggtaa cccaatccca gtacatgttt 6660
gggttcaaca aattagtttg tggatctctt ctttgagcgt gcatcaaaga tgggaacttg 6720
actggatcac gaatgaagaa gaatgggaaa ttcaacatga cccaatccca attaccttca 6780
gtagtgtaga acttacaagc catacctttc aaatctctca taccttcgtt agaacctctt 6840
tccaaaccag tggtagagaa tctagtaaca catggagtct ttttaccgac accaaccaac 6900
atatcgatat cgcaaatatc ggagatatcg gcagtaactt caaattcacc ataagcacca 6960
gcacctttag cgtgaacaac tctttctgga atcttttctc tgttgaaatg ggccaaggta 7020
tcaaccaaat gatgatcaga caaagccaaa gttctaccat cagctggtgg accttcaggc 7080
ttatgaaatg gacaaccatt agcagtagtg taagttggct tagaatctgg agccatttta 7140
agctttgttt tatatttgtt gtaaaaagta gataattact tccttgatga tctgtaaaaa 7200
agagaaaaag aaagcatcta agaacttgaa aaactacgaa ttagaaaaga ccaaatatgt 7260
atttcttgca ttgaccaatt tatgcaagtt tatatatatg taaatgtaag tttcacgagg 7320
ttctactaaa ctaaaccacc cccttggtta gaagaaaaga gtgtgtgaga acaggctgtt 7380
gttgtcacac gattcggaca attctgtttg aaagagagag agtaacagta cgatcgaacg 7440
aactttgctc tggagatcac agtgggcatc atagcatgtg gtactaaacc ctttcccgcc 7500
attccagaac cttcgattgc ttgttacaaa acctgtgagc cgtcgctagg accttgttgt 7560
gtgacgaaat tggaagctgc aatcaatagg aagacaggaa gtcgagcgtg tctgggtttt 7620
ttcagttttg ttctttttgc aaacaaatca cgagcgacgg taatttcttt ctcgataaga 7680
ggccacgtgc tttatgaggg taacatcaat tcaagaagga gggaaacact tcctttttct 7740
ggccctgata atagtatgag ggtgaagcca aaataaagga ttcgcgccca aatcggcatc 7800
tttaaatgca ggtatgcgat agttcctcac tctttcctta ctcacgagta attcttgcaa 7860
atgcctatta tgcagatgtt ataatatctg tgcgtacgcg cccagttcga gtttatcatt 7920
atcaatactg ccatttcaaa gaatacgtaa ataattaata gtagtgattt tcctaacttt 7980
atttagtcaa aaaattagcc ttttaattct gctgtaaccc gtacatgccc aaaatagggg 8040
gcgggttaca cagaatatat aacatcgtag gtgtctgggt gaacagttta ttcctggcat 8100
ccactaaata taatggagcc cgctttttaa gctggcatcc agaaaaaaaa agaatcccag 8160
caccaaaata ttgttttctt caccaaccat cagttcatag gtccattctc ttagcgcaac 8220
tacagagaac aggggcacaa acaggcaaaa aacggacaca acctcaatgg agtgatgcaa 8280
cctgcctgga gtaaatgatg acacaaggca attgacccac gcatgtatct atctcatttt 8340
cttacacctt ctattacctt ctgctctctc tgatttggaa aaagctgaaa aaaaaggttg 8400
aaaccagttc cctgaaatta ttcccctact tgactaataa gtatataaag acggtaggta 8460
ttgattgtaa ttctgtaaat ctatttctta aacttcttaa attctacttt tatagttagt 8520
ctttttttta gttttaaaac accaagaact tagtttcgaa taaacacaca taaacaaaca 8580
aaaagcttaa aatgaccatt accgaaactt ctaagccaac cttgagaaga attccaagat 8640
cagctggtga tgaagctatt ttccaagtct tgcaagaaga tggcgttgtt gttatcgaag 8700
gtttcatgtc tgctgaccaa gttagaagat tcaacggtga aattgatcca cacatgaagc 8760
aatgggaatt gggtcaaaaa tcctaccaag aatcttactt ggctggtatg agacaattgt 8820
catctttgcc tttgttctcc aagttgttca gagatgaatt gatgaacgac gaattattgc 8880
acggtttgtg caagagatta ttcggtccag aatctggtga ttactggttg actacttctt 8940
ctgttttgga aactgaacca ggttaccatg gtcaagaatt gcatagagaa catgatggta 9000
ttccaatctg tactaccttg ggtagacaat ctccagaatc tatgttgaac ttcttgactg 9060
ctttgactga tttcactgct gaaaatggtg ctactagagt tttgccaggt tctcatttgt 9120
gggaagattt ttctgctcca ccaccaaaag ctgatactgc tattccagct gttatgaatc 9180
caggtgatgc tgttttgttt actggtaaaa ctttacatgg tgccggtaag aacaacacta 9240
ctgatttttt gagaagaggt ttcccattga tcatgcaatc ttgtcaattc actccagttg 9300
aagctagtgt tgctttgcca agagaattgg ttgaaactat gactccattg gcccaaaaaa 9360
tggttggttg gagaactgtt tctgctaagg gtgttgatat ttggacctac gatttgaagg 9420
atttggctac cggtattgac ttgaagtcta atcaagttgc taagaaggcc taaccgcggc 9480
acgtccgacg gcggcccgac gggtccgagg cctcggagat ccgtccccct tttcctttgt 9540
cgatatcatg taattagtta tgtcacgctt acattcacgc cctcccccca catccgctct 9600
aaccgaaaag gaaggagtta gacaacctga agtctaggtc cctatttatt tttttatagt 9660
tatgttagta ttaagaacgt tatttatatt tcaaattttt cttttttttc tgtacagacg 9720
cgcctgccta cgcaacactt tagctgcttt ttttaagcag cggcagatga ggatttaaat 9780
aacgcataac caagaatata cctaatgaag tgaaggtata caggctgttc tatggacttg 9840
aataatactg gctaccctgg cgtcctaatg tcgttaagaa agtaacttga tatcagatta 9900
atcacaaact tggtccacaa aataccactt atctcatata atctaaatct gactaaaggg 9960
tgtaggcacg gaacttcgga cctagaggta ttgctcaata tgtatatttc tcaatggttc 10020
atctaggtaa gcaaggcctt gaataatata cttaatcttg tctgtttcgc aacaattgct 10080
gagtacaata atgttggaat aaaaatcaac tatcatctac taactagtat ttacgttact 10140
agtatattat catatacggt gttagaagat gacgcaaatg atgagaaata gtcatctaaa 10200
ttagtggaag ctgaaacgca aggattgata atgtaatagg atcaatgaat attaacatat 10260
aaaatgatga taataatatt tatagaattg tgtagaattg cagattccct tttatggatt 10320
cctaaatcct cgaggagaac ttctagtata tctacatacc taatattata gccttaatca 10380
caatggaatc ccaacaacct gcagggcatg caagcttggc gtaatcatgg tcatagctgt 10440
ttcctgtgtg aaattgttat ccgctcacaa ttccacacaa catacgagcc ggaagcataa 10500
agtgtaaagc ctggggtgcc taatgagtga gctaactcac attaattgcg ttgcgctcac 10560
tgcccgcttt ccagtcggga aacctgtcgt gccagctgca ttaatgaatc ggccaacgcg 10620
cggggagagg cggtttgcgt attgggcgct cttccgcttc ctcgctcact gactcgctgc 10680
gctcggtcgt tcggctgcgg cgagcggtat cagctcactc aaaggcggta atacggttat 10740
ccacagaatc aggggataac gcaggaaaga acatgtgagc aaaaggccag caaaaggcca 10800
ggaaccgtaa aaaggccgcg ttgctggcgt ttttccatag gctccgcccc cctgacgagc 10860
atcacaaaaa tcgacgctca agtcagaggt ggcgaaaccc gacaggacta taaagatacc 10920
aggcgtttcc ccctggaagc tccctcgtgc gctctcctgt tccgaccctg ccgcttaccg 10980
gatacctgtc cgcctttctc ccttcgggaa gcgtggcgct ttctcatagc tcacgctgta 11040
ggtatctcag ttcggtgtag gtcgttcgct ccaagctggg ctgtgtgcac gaaccccccg 11100
ttcagcccga ccgctgcgcc ttatccggta actatcgtct tgagtccaac ccggtaagac 11160
acgacttatc gccactggca gcagccactg gtaacaggat tagcagagcg aggtatgtag 11220
gcggtgctac agagttcttg aagtggtggc ctaactacgg ctacactaga aggacagtat 11280
ttggtatctg cgctctgctg aagccagtta ccttcggaaa aagagttggt agctcttgat 11340
ccggcaaaca aaccaccgct ggtagcggtg gtttttttgt ttgcaagcag cagattacgc 11400
gcagaaaaaa aggatctcaa gaagatcctt tgatcttttc tacggggtct gacgctcagt 11460
ggaacgaaaa ctcacgttaa gggattttgg tcatgagatt atcaaaaagg atcttcacct 11520
agatcctttt aaattaaaaa tgaagtttta aatcaatcta aagtatatat gagtaaactt 11580
ggtctgacag ttaccaatgc ttaatcagtg aggcacctat ctcagcgatc tgtctatttc 11640
gttcatccat agttgcctga ctccccgtcg tgtagataac tacgatacgg gagggcttac 11700
catctggccc cagtgctgca atgataccgc gagacccacg ctcaccggct ccagatttat 11760
cagcaataaa ccagccagcc ggaagggccg agcgcagaag tggtcctgca actttatccg 11820
cctccatcca gtctattaat tgttgccggg aagctagagt aagtagttcg ccagttaata 11880
gtttgcgcaa cgttgttgcc attgctacag gcatcgtggt gtcacgctcg tcgtttggta 11940
tggcttcatt cagctccggt tcccaacgat caaggcgagt tacatgatcc cccatgttgt 12000
gcaaaaaagc ggttagctcc ttcggtcctc cgatcgttgt cagaagtaag ttggccgcag 12060
tgttatcact catggttatg gcagcactgc ataattctct tactgtcatg ccatccgtaa 12120
gatgcttttc tgtgactggt gagtactcaa ccaagtcatt ctgagaatag tgtatgcggc 12180
gaccgagttg ctcttgcccg gcgtcaatac gggataatac cgcgccacat agcagaactt 12240
taaaagtgct catcattgga aaacgttctt cggggcgaaa actctcaagg atcttaccgc 12300
tgttgagatc cagttcgatg taacccactc gtgcacccaa ctgatcttca gcatctttta 12360
ctttcaccag cgtttctggg tgagcaaaaa caggaaggca aaatgccgca aaaaagggaa 12420
taagggcgac acggaaatgt tgaatactca tactcttcct ttttcaatat tattgaagca 12480
tttatcaggg ttattgtctc atgagcggat acatatttga atgtatttag aaaaataaac 12540
aaataggggt tccgcgcaca tttccccgaa aagtgccacc tgacgtctaa gaaaccatta 12600
ttatcatgac attaacctat aaaaataggc gtatcacgag gccctttcgt c 12651
<210> 172
<211> 1850
<212> DNA
<213> Artificial Sequence
<220>
<223> PF cassette including both AscI sites
<400> 172
ggcgcgccat gcagggatat cagatctatg cgactgggtg agcatatgtt ccgctgatgt 60
gatgtgcaag ataaacaagc aaggcagaaa ctaacttctt cttcatgtaa taaacacacc 120
ccgcgtttat ttacctatct ctaaacttca acaccttata tcataactaa tatttcttga 180
gataagcaca ctgcacccat accttcctta aaaacgtagc ttccagtttt tggtggttcc 240
ggcttccttc ccgattccgc ccgctaaacg catatttttg ttgcctggtg gcatttgcaa 300
aatgcataac ctatgcattt aaaagattat gtatgctctt ctgacttttc gtgtgatgag 360
gctcgtggaa aaaatgaata atttatgaat ttgagaacaa ttttgtgttg ttacggtatt 420
ttactatgga ataatcaatc aattgaggat tttatgcaaa tatcgtttga atatttttcc 480
gaccctttga gtacttttct tcataattgc ataatattgt ccgctgcccc tttttctgtt 540
agacggtgtc ttgatctact tgctatcgtt caacaccacc ttattttcta actatttttt 600
ttttagctca tttgaatcag cttatggtga tggcacattt ttgcataaac ctagctgtcc 660
tcgttgaaca taggaaaaaa aaatatataa acaaggctct ttcactctcc ttgcaatcag 720
atttgggttt gttcccttta ttttcatatt tcttgtcata ttcctttctc aattattatt 780
ttctactcat aacctcacgc aaaataacac agtcaaatct atcaaaaagc ttggcctgca 840
gggccagctt acccttaaat ttatttgcac tactggaaaa ctacctgttc catggccaac 900
acttgtcact actttctctt atggtgttca atgcttttcc cgttatccgg atcatatgaa 960
acggcatgac tttttcaaga gtgccatgcc cgaaggttat gtacaggaac gcactatatc 1020
tttcaaagat gacgggaact acaagacgcg tgctgaagtc aagtttgaag gtgataccct 1080
tgttaatcgt atcgagttaa aaggtattga ttttaaagaa gatggaaaca ttctcggaca 1140
caaactcgag tacaactata actcacacaa tgtatacatc acggcagaca aacaaaagaa 1200
tggaatcaaa gctaacttca aaattcgcca caacattgaa gatggatccg ttcaactagc 1260
agaccattat caacaaaata ctccaattgg cgatggccct gtccttttac cagacaacca 1320
ttacctgtcg acacaatctg ccctttcgaa agatcccaac gaaaagcgtg accacatggt 1380
ccttcttgag tttgtaactg ctgctgggat tacacatggc atggatgagc tctacaaata 1440
atgaattcgt cgaggccgtt caggcctcga ggccgttcag gctcgacccg gggatccgcg 1500
gttaattcaa attaattgat atagtttttt aatgagtatt gaatctgttt agaaataatg 1560
gaatattatt tttatttatt tatttatatt attggtcggc tcttttcttc tgaaggtcaa 1620
tgacaaaatg atatgaagga aataatgatt tctaaaattt tacaacgtaa gatattttta 1680
caaaagccta gctcatcttt tgtcatgcac tattttactc acgcttgaaa ttaacggcca 1740
gtccactgcg gagtcatttc aaagtcatcc taatcgatct atcgtttttg atagctcatt 1800
ttggagcatg cccatgggtt aactgatcaa tgcatcctgc atggcgcgcc 1850
<210> 173
<211> 15212
<212> DNA
<213> Artificial Sequence
<220>
<223> HRT1 containing DmaW, EasF, EasC, and EasE
<400> 173
accacggtga acaatccccg ctggctcata tttgccgccg gttcccgtaa atcctccggt 60
acgctccagc gtgttttatc tctgcgagca taatgcctgc gtcatccgcc agcaggagct 120
ggactttact gatgcccgtt atatctgcga aaagaccggg atctggaccc gtgatggcat 180
tctctggttt tcgtcatccg gtgaagagat tgagccacct gacagtgtga cctttcacat 240
ctggacagcg tacagcccgt tcaccacctg ggtgcagatt gtcaaagact ggatgaaaac 300
gaaaggggat acgggaaaac gtaaaacctt cgtaaacacc acgctcggtg agacgtggga 360
ggcgaaaatt ggcgaacgtc cggatgctga agtgatggca gagcggaaag agcattattc 420
agcgcccgtt cctgaccgtg tggcttacct gaccgccggt atcgactccc agctggaccg 480
ctacgaaatg cgcgtatggg gatgggggcc gggtgaggaa agctggctga ttgaccggca 540
gattattatg ggccgccacg acgatgaaca gacgctgctg cgtgtggatg aggccatcaa 600
taaaacctat acccgccgga atggtgcaga aatgtcgata tcccgtatct gctgggatac 660
tggcgttttc ccgtctttca gtgccttgtt cagttcttcc tgacgggcgg tatatttctc 720
cagcttacgc gccatgcagg gatatcagat cttcgaggag aacttctagt atatccacat 780
acctaatatt attgccttat taaaaatgga atcccaacaa ttacatcaaa atccacattc 840
tcttcaaaat caattgtcct gtacttcctt gttcatgtgt gttcaaaaac gttatattta 900
taggataatt atactctatt tctcaacaag taattggttg tttggccgag cggtctaagg 960
cgcctgattc aagaaatatc ttgaccgcag ttaactgtgg gaatactcag gtatcgtaag 1020
atgcaagagt tcgaatctct tagcaaccat tatttttttc ctcaacataa cgagaacaca 1080
caggggcgct atcgcacaga atcaaattcg atgactggaa attttttgtt aatttcagag 1140
gtcgcctgac gcatatacct ttttcaactg aaaaattggg agaaaaagga aaggtgagag 1200
gccggaaccg gcttttcata tagaatagag aagcgttcat gactaaatgc ttgcatcaca 1260
atacttgaag ttgacaatat tatttaagga cctattgttt tttccaatag gtggttagca 1320
atcgtcttac tttctaactt ttcttacctt ttacatttca gcaatatata tatatatttc 1380
aaggatatac cattctaatg tctgccccta tgtctgcccc taagaagatc gtcgttttgc 1440
caggtgacca cgttggtcaa gaaatcacag ccgaagccat taaggttctt aaagctattt 1500
ctgatgttcg ttccaatgtc aagttcgatt tcgaaaatca tttaattggt ggtgctgcta 1560
tcgatgctac aggtgtccca cttccagatg aggcgctgga agcctccaag aaggttgatg 1620
ccgttttgtt aggtgctgtg gctggtccta aatggggtac cggtagtgtt agacctgaac 1680
aaggtttact aaaaatccgt aaagaacttc aattgtacgc caacttaaga ccatgtaact 1740
ttgcatccga ctctctttta gacttatctc caatcaagcc acaatttgct aaaggtactg 1800
acttcgttgt tgtcagagaa ttagtgggag gtatttactt tggtaagaga aaggaagacg 1860
atggtgatgg tgtcgcttgg gatagtgaac aatacaccgt tccagaagtg caaagaatca 1920
caagaatggc cgctttcatg gccctacaac atgagccacc attgcctatt tggtccttgg 1980
ataaagctaa tcttttggcc tcttcaagat tatggagaaa aactgtggag gaaaccatca 2040
agaacgaatt ccctacattg aaggttcaac atcaattgat tgattctgcc gccatgatcc 2100
tagttaagaa cccaacccac ctaaatggta ttataatcac cagcaacatg tttggtgata 2160
tcatctccga tgaagcctcc gttatcccag gttccttggg tttgttgcca tctgcgtcct 2220
tggcctcttt gccagacaag aacaccgcat ttggtttgta cgaaccatgc cacggttctg 2280
ctccagattt gccaaagaat aaggttgacc ctatcgccac tatcttgtct gctgcaatga 2340
tgttgaaatt gtcattgaac ttgcctgaag aaggtaaggc cattgaagat gcagttaaaa 2400
aggttttgga tgcaggtatc agaactggtg atttaggtgg ttccaacagt accaccgaag 2460
tcggtgatgc tgtcgccgaa gaagttaaga aaatccttgc ttaaaaagat tctctttttt 2520
tatgatattt gtacataaac tttataaatg aaattcataa tagaaacgac acgaaattac 2580
aaaatggaat atgttcatag ggtagacgaa actatatacg caatctacat acatttatca 2640
agaaggagaa aaaggaggat agtaaaggaa tacaggtaag caaattgata ctaatggctc 2700
aacgtgataa ggaaaaagaa ttgcacttta acattaatat tgacaaggag gagggcacca 2760
cacaaaaagt taggtgtaac agaaaatcat gaaactacga ttcctaattt gatattggag 2820
gattttctct aaaaaaaaaa aaatacaaca aataaaaaac actcaatgac ctgaccattt 2880
gatggagttt aagtcaatac cttcttgaag catttcccat aatggtgaaa gttccctcaa 2940
gaattttact ctgtcagaaa cggccttacg acgtagtcga gcatgcgtat tgggcgctct 3000
tccgcttcct cgctcactga ctcgctgcgc tcggtcgttc ggctgcggcg agcggtatca 3060
gctcactcaa aggcggtaat acggttatcc acagaatcag gggataacgc aggaaagaac 3120
atgtgagcaa aaggccagca aaaggccagg aaccgtaaaa aggccgcgtt gctggcgttt 3180
ttccataggc tccgcccccc tgacgagcat cacaaaaatc gacgctcaag tcagaggtgg 3240
cgaaacccga caggactata aagataccag gcgtttcccc ctggaagctc cctcgtgcgc 3300
tctcctgttc cgaccctgcc gcttaccgga tacctgtccg cctttctccc ttcgggaagc 3360
gtggcgcttt ctcatagctc acgctgtagg tatctcagtt cggtgtaggt cgttcgctcc 3420
aagctgggct gtgtgcacga accccccgtt cagcccgacc gctgcgcctt atccggtaac 3480
tatcgtcttg agtccaaccc ggtaagacac gacttatcgc cactggcagc agccactggt 3540
aacaggatta gcagagcgag gtatgtaggc ggtgctacag agttcttgaa gtggtggcct 3600
aactacggct acactagaag gacagtattt ggtatctgcg ctctgctgaa gccagttacc 3660
ttcggaaaaa gagttggtag ctcttgatcc ggcaaacaaa ccaccgctgg tagcggtggt 3720
ttttttgttt gcaagcagca gattacgcgc agaaaaaaag gatctcaaga agatcctttg 3780
atcttttcta cggggtctga cgctcagtgg aacgaaaact cacgttaagg gattttggtc 3840
atgagattat caaaaaggat cttcacctag atccttttaa attaaaaatg aagttttaaa 3900
tcaatctaaa gtatatatga gtaaacttgg tctgacagtt aacggcgcgt tcatcgtcca 3960
cctccggaga acaggccacc atcacgcatc tgtgtctgaa tttcatcacg acgcgcctta 4020
agggcaccaa taactgcctt aaaaaaatta cgccccgccc tgccactcat cgcagtactg 4080
ttgtaattca ttaagcattc tgccgacatg gaagccatca cagacggcat gatgaacctg 4140
aatcgccagc ggcatcagca ccttgtcgcc ttgcgtataa tatttgccca tggtgaaaac 4200
gggggcgaag aagttgtcca tattggccac gtttaaatca aaactggtga aactcaccca 4260
gggattggct gagacgaaaa acatattctc aataaaccct ttagggaaat aggccaggtt 4320
ttcaccgtaa cacgccacat cttgcgaata tatgtgtaga aactgccgga aatcgtcgtg 4380
gtattcactc cagagcgatg aaaacgtttc agtttgctca tggaaaacgg tgtaacaagg 4440
gtgaacacta tcccatatca ccagctcacc gtctttcatt gccatacgga attccggatg 4500
agcattcatc aggcgggcaa gaatgtgaat aaaggccgga taaaacttgt gcttattttt 4560
ctttacggtc tttaaaaagg ccgtaatatc cagctgaacg gtctggttat aggtacattg 4620
agcaactgac tgaaatgcct caaaatgttc tttacgatgc cattgggata tatcaacggt 4680
ggtatatcca gtgatttttt tctccatttt agcttcctta gctcctgaaa atctcgataa 4740
ctcaaaaaat acgcccggta gtgatcttat ttcattatgg tgaaagttgg aacctcttac 4800
gtgccgatca acgtctcatt ttcgccaaaa gttggcccag ggcttcccgg tatcaacagg 4860
gacaccagga tttatttatt ctgcgaagtg atcttccgtc acaggtattg gaccaccctg 4920
tgggtttata agcgcgctgc tggcgtgtaa ggcggtgacg gcgaaggaag ggtccttttc 4980
atcacgtgct ataaaaataa ttataattta aattttttaa tataaatata taaattaaaa 5040
atagaaagta aaaaaagaaa ttaaagaaaa aatagttttt gttttccgaa gatgtaaaag 5100
actctagggg gatcgccaac aaatactacc ttttatcttg ctcttcctgc tctcaggtat 5160
taatgccgaa ttgtttcatc ttgtctgtgt agaagaccac acacgaaaat cctgtgattt 5220
tacattttac ttatcgttaa tcgaatgtat atctatttaa tctgcttttc ttgtctaata 5280
aatatatatg taaagtacgc tttttgttga aattttttaa acctttgttt attttttttt 5340
cttcattccg taactcttct accttcttta tttactttct aaaatccaaa tacaaaacat 5400
aaaaataaat aaacacagag taaattccca aattattcca tcattaaaag atacgaggcg 5460
cgtgtaagtt acaggcaagc gatccgtcct aagaaaccat tattatcatg acattaacct 5520
ataaaaatag gcgtatcacg aggccctttc gtctcgcgcg tttcggtgat gacggtgaaa 5580
acctctgaca catgcagctc ccggagacgg tcacagcttg tctgtaagcg gatgccggga 5640
gcagacaagc ccgtcagggc gcgtcagcgg gtgttggcgg gtgtcggggc tggcttaact 5700
atgcggcatc agagcagatt gtactgagag tgcaccacgg cgcgtggcac ccttgcgggc 5760
catgtcatac accgccttca gagcagccgg acctatctgc ccgttacgcg ccagcttgca 5820
aattaaagcc ttcgagcgtc ccaaaacctt ctcaagcaag gttttcagta taatgttaca 5880
tgcgtacacg cgtctgtaca gaaaaaaaag aaaaatttga aatataaata acgttcttaa 5940
tactaacata actataaaaa aataaatagg gacctagact tcaggttgtc taactccttc 6000
cttttcggtt agagcggatg tggggggagg gcgtgaatgt aagcgtgaca taactaatta 6060
catgatatcg acaaaggaaa agggggacgg atctccgagg cctcggaccc gtcgggccgc 6120
cgtcggacgt gccgcggtta tggagttgca gcaccaacgt taatattggc accgtaaact 6180
ggtctagaat cttcagacaa caaccagacg atagttctag caacatcttc aggttgaatg 6240
attgggaaac cttctttgat gtaggcttct tttacttcgt cgatagattt agcttgtggc 6300
atgaattgtg gcaaagctgg agtatcggta atacctggag aaacagaatt gattctaata 6360
cccaatggcc aagcatccat agcagcacaa gaagtaaagt aagcacaagc acctttagaa 6420
gtaccgtaag cataagcatc tggcaaatgg ataaaggcag caatggaacc aatgttaacg 6480
atagctctat cttttggacc tggcaaagcc ttcatagctc taataccagc tctagtacag 6540
tagaaaacac cgtccaagtt aaccttgaaa actctttgcc attcttcatc agtttcttcc 6600
aacaaagctg gagcttgtct agcaccagct ggttgagcaa taccagcaac attagccaaa 6660
ccatgcaagt caccaaaagt ttgaacgata gtttgcaacc aagcatcaac ttcagcggat 6720
gaagaaacat ccaatctagt gcaatgaacc tttgtagctg gattcaaggc ttgaatatca 6780
gaggtaacgg attcgaaacc ctttggagaa atatcaccaa tgcatataac agcagcacct 6840
ctagcagcca ataatctaca agtagcagca ccaataccag aagcaccacc agtaacagcg 6900
aaaattctag agttgacaac ggtcataagc tttttgtttg tttatgtgtg tttattcgaa 6960
actaagttct tggtgtttta aaactaaaaa aaagactaac tataaaagta gaatttaaga 7020
agtttaagaa atagatttac agaattacaa tcaataccta ccgtctttat atacttatta 7080
gtcaagtagg ggaataattt cagggaactg gtttcaacct tttttttcag ctttttccaa 7140
atcagagaga gcagaaggta atagaaggtg taagaaaatg agatagatac atgcgtgggt 7200
caattgcctt gtgtcatcat ttactccagg caggttgcat cactccattg aggttgtgtc 7260
cgttttttgc ctgtttgtgc ccctgttctc tgtagttgcg ctaagagaat ggacctatga 7320
actgatggtt ggtgaagaaa acaatatttt ggtgctggga ttcttttttt ttctggatgc 7380
cagcttaaaa agcgggctcc attatattta gtggatgcca ggaataaact gttcacccag 7440
acacctacga tgttatatat tctgtgtaac ccgcccccta ttttgggcat gtacgggtta 7500
cagcagaatt aaaaggctaa ttttttgact aaataaagtt aggaaaatca ctactattaa 7560
ttatttacgt attctttgaa atggcagtat tgataatgat aaactcgaac tgggcgcgtc 7620
gtgccgtcgt tgttaatcac cacatggtta ttctgctcaa acgtcccgga cgcctgcgaa 7680
cgcgccgaag gaaaatgaga aatatcgagg gagacgattc agaggagcag gacaaactat 7740
aaccgactgt ttgttggagg atgccgtaca taacgaacac tgctgaagct accatgtcta 7800
cagtttagag gaatgggtac aactcacagg cgagggatgg tgttcactcg tgctagcaaa 7860
cgcggtggga gcaaaaagta gaatattatc ttttattcgt gaaacttcga acactgtcat 7920
ctaaagatgc tatatactaa tataggcata cttgataatg aaaactataa atcgtaaaga 7980
cataagagat ccgcggttat ggactttttc tgttcctcta acgcttccaa caggaggata 8040
accttatcca tactggcaga acccatatcg attactaagc tatcttgagc gattgaagca 8100
gccatcttac tagcttcagc tttgaggatg catagttcct catgtctagg atagaaatca 8160
gcagcgtggg aatgtctatt ccaatgctga atgccttttg ttgagtaaaa caaaagtgca 8220
ggaagtgtct tgacttcctt agttagacct tcgacaactt ggtctggtat agaactttcc 8280
aaccctgctt gtctaatatc aataataggt ggagcagaaa ttgtcattta gttcaatctt 8340
agtctcaatt gatagaatgc tacgtcacag tctggtgctg tccacactgc ggcatcctcc 8400
aaacccgctg aatgaaacaa ctgatccctt tcagctttgt cgtatttgta agagcatcca 8460
aatctgatct tttcatcacg tctgactata acagtctcta gtggtgcacc taattggata 8520
tcttttgatt gtgtgatcaa actagcttcg tgacgaccca atgcatggtt ccattcagaa 8580
tgaaagtacc aatcctcttc attgaaaatc tttctgtcgc cttttggatg aaacaatgaa 8640
ttcgcatagc ttagtgaggc aagagcaaat ggaactactc catctgcggt gtaggctctt 8700
aagattttcg taggaagttt acatgaatct agagacacta taatggatga ttgagttggg 8760
gatgtagaca gagcagattg ggcgatattt ctgaggaaag atgcagcatt atctcgtgag 8820
aagttaccga tggttaaccc aaataacagt atacatcttg gtctgttcct aatatcagga 8880
gcgttttgta accagtgaag tccatcgtca aaagtgccat gtaaggctgc gaatctaaca 8940
tagtggaatc tgtccactgg aatggcttga aaattagatg ctaattcaga gtaggacagg 9000
tccaaggcgt agtaagaagc tttgttttat atttgttgta aaaagtagat aattacttcc 9060
ttgatgatct gtaaaaaaga gaaaaagaaa gcatctaaga acttgaaaaa ctacgaatta 9120
gaaaagacca aatatgtatt tcttgcattg accaatttat gcaagtttat atatatgtaa 9180
atgtaagttt cacgaggttc tactaaacta aaccaccccc ttggttagaa gaaaagagtg 9240
tgtgagaaca ggctgttgtt gtcacacgat tcggacaatt ctgtttgaaa gagagagagt 9300
aacagtacga tcgaacgaac tttgctctgg agatcacagt gggcatcata gcatgtggta 9360
ctaaaccctt tcccgccatt ccagaacctt cgattgcttg ttacaaaacc tgtgagccgt 9420
cgctaggacc ttgttgtgtg acgaaattgg aagctgcaat caataggaag acaggaagtc 9480
gagcgtgtct gggttttttc agttttgttc tttttgcaaa caaatcacga gcgacggtaa 9540
tttctttctc gataagaggc cacgtgcttt atgagggtaa catcaattca agaaggaggg 9600
aaacacttcc tttttctggc cctgataata gtatgagggt gaagccaaaa taaaggattc 9660
gcgcccaaat cggcatcttt aaatgcaggt atgcgatagt tcctcactct ttccttactc 9720
acgagtaatt cttgcaaatg cctattatgc agatgttata atatctgtgc gtggcgcgtc 9780
cggctgtctg ccatgctgcc cggtgtaccg acataaccgc cggtggcata gccgcgcata 9840
cgcgccattt ccttccatct tgtgattcat gctatccatc ttttttgagt atccaattaa 9900
cgaagacgtt accagctgat tgaaggttct caaagtgact gtactccatg ttttcttatc 9960
atccatgtag ttatttttca aactgcaaat tcaagaaaaa gccacgcgtg tgcacctttt 10020
ttttcccctt ccagtgcatt atgcaataga cagcacgagt ctttgaaaaa gtaacttata 10080
aaactgtatc aatttttaaa cctaaataga ttcataaact attcgttaat ataaagtgtt 10140
ctaaactatg atgaaaaaat aagcagaaaa gactaataat tcttagttaa aagcactccg 10200
cggtcaaaca acggaagcag acaaagaacc caagaaagat tgaacccact taacagcaac 10260
attccaagca ccttgtcttg tttgtctaca cataccttca gcatcccaag catcaccatc 10320
aatacccaat ttggagaaga acaaaccttc tggatcccat cttctcttaa cttcaaccaa 10380
tctagcgtag ttagaacccc aataaacttc tctggatctt ctttcggatg gatcacccaa 10440
atttctgtag gagatttgga aggctggttc aatttcgtat aatctggcca tttgaacggt 10500
ttgcaattca tgtaaagcag tagctttagc agccaaagat ggagcaccaa cacctctaac 10560
gaaattcaat aattgagcag catctctcca acctggatgc atagcagtag aagctggatc 10620
tctattcaat gaagagactc taccacccaa gttagaagtg aacaacaaat catctggagt 10680
cattggcaat ctagcaaaat gttgtgccaa agcagctgga ccagtagcag aattaaacaa 10740
gtccaaggaa accaaaacgg aacccatcaa aataccgtaa tcttctgggt atctatcagc 10800
ggtatgtctg aaagtagaag agatttgtgg caagaatcta gaggtcaatt ggtatggatg 10860
accattatgt tccaaagctc ttctcaaagg atccattctt tcgtcagcaa cagctggaac 10920
agtcaagtta gagaagtaca tagtcaaact agcttggacg gtatcatcat cagtgtttct 10980
caaaatgaat tgaccggcag taccatggtt gttcaagaat tgtaaagctc tcaacaaagc 11040
agcaacacca tgtgaccaaa aagtagcatc agctctagaa ccagtaacaa cagcatcagc 11100
aacagaaact ggcaaatctg gatgaacttg caatgtagct tgagtaacaa caccaaaagt 11160
accaccacca ccgcctctca aagcccaaaa caaatctggg ttttgatgag cgttagcaat 11220
aacgatttca cccttagcag taacaacttc gtattccaag acgttatcaa cagccaaacc 11280
atgagcataa gacaaccaag aagaaacacc accaccttgt aaaaaaccac cagcagcacc 11340
aactgttgga cattcaccac caacaactat ccaaccttca gaagcacctc tagcatacaa 11400
gtcacccaac aaaataccag caccaacagt aacagctggt tgacctggag cagattgtct 11460
ttcagaggaa ccagctggtc taaaatttgg atggtattga acggagttca agtgatgggt 11520
atgaatttca acagaatctg gaccagatga tctaccagaa ccatcatgac cggtgtttct 11580
aataaccaat ctcaagttgt gttgtctggt gaaatcaaca accttttgaa catcagaagc 11640
ggacttaaca gcagcggaat ataatggaat tctaccttga tgacaagaac cagctggacc 11700
tgatctcaag tagtaacaag tttgattaga ttcagcacca ccttcccaaa cccaatcttg 11760
taaagcacca gtatgagaag cacgccactt agaatccaag gattcttgta aaacttgttc 11820
gcaagcagaa gaatcttgac cggattgatg acaaacatga gcaattggct taacatgagc 11880
caaagcacca ccaatagatt cgttcaaagt agaccaatga gaaacagttg gccaacatgg 11940
ttcacctggt ctacatctgc aatcatgtct atcatcatca acagcagaca atctagacaa 12000
gtaggcacca accaaaataa ccaagatcaa ttgtctaaca ccacccaaaa tacctctgga 12060
ttgacccata agctttttgt aattaaaact tagattagat tgctatgctt tctttctaat 12120
gagcaagaag taaaaaaagt tgtaatagaa caagaaaaat gaaactgaaa cttgagaaat 12180
tgaagaccgt ttattaactt aaatatcaat gggaggtcat cgaaagagaa aaaaatcaaa 12240
aaaaaaaatt ttcaagaaaa agaaacgtga taaaaatttt tattgccttt ttcgacgaag 12300
aaaaagaaac gaggcggtct cttttttctt ttccaaacct ttagtacggg taattaacga 12360
caccctagag gaagaaagag gggaaattta gtatgctgtg cttgggtgtt ttgaagtggt 12420
acggcgatgc gcggagtccg agaaaatctg gaagagtaaa aaaggagtag aaacattttg 12480
aagctaggcg cgtcagccgg taaagattcc ccacgccaat ccggctggtt gcctccttcg 12540
tgaagacaaa ctcacgcgcc tccaaaatga gctatcaaaa acgatagatc gattaggatg 12600
actttgaaat gactccgcag tggactggcc gttaatttca agcgtgagta aaatagtgca 12660
tgacaaaaga tgagctaggc ttttgtaaaa atatcttacg ttgtaaaatt ttagaaatca 12720
ttatttcctt catatcattt tgtcattgac cttcagaaga aaagagccga ccaataatat 12780
aaataaataa ataaaaataa tattccatta tttctaaaca gattcaatac tcattaaaaa 12840
actatatcaa ttaatttgaa ttaaccgcgg tcacaatcta gctggaataa aagattcttc 12900
agctggagct tcagactttt gcaacaaggt ttcaacacct ttttgaattc tagcagccaa 12960
agctggagaa accaacttcc aggtttcgta aacttgcttt tgaatttcgg ctctggtttg 13020
agcaacagat tcaaccatgt tttggaccat ggtatcttgg aattcttgac ctctaaaatc 13080
tggcaaaact tcgtagaaat ctctggcgaa cttgtaatca tgttcagtag tttgcaacca 13140
ggacttggat tggatttctt tcaaccattc attatacttt ggatctggtt ctctaggttc 13200
ggcaaatcta actggttgtt gttgagtcaa gaaacctgga tgagcaccat aattagcatc 13260
aaaagtacct tgaccatctc tagcaattgg gttaaaggta tgagcggatc tattaactgg 13320
gatttgttga tggttagcac ccaatctata tctttgagcg tctgggtaag cgaaaattct 13380
agcttgtaaa attggatctt cagatggagc aacacctgga accatgttag atggagagta 13440
agccaattgt tcaatttcgg cgaagtagtt agctgggttt cttttcaaag tcaacttacc 13500
gaaaaccttt ggtgggatca aagtaacatc ttttgggtaa gtacccaagt tccagtgctt 13560
agtaacatcc aaaatgttgt aacccaagtt tggagcatct tttggatcaa caacttgaac 13620
agaagcagtc caagttgggt attcacctct ttcaatagct tcatacaagt ctctagtagc 13680
gtgatctgga tccaaatcat ttgtaccttt agcttgttga ccatcggtga aatttggacc 13740
tctatcagat gacaagaaga tgtggacgta cttaaaagaa ccatctggct taacccattt 13800
gtaagcatga gccatataac cagacatgga tctccaattg aacatagtac cgaaatcaga 13860
aaattgcaac aagaccatgt gcaaggattc atgattgttg gtaacccaat cccagtacat 13920
gtttgggttc aacaaattag tttgtggatc tcttctttga gcgtgcatca aagatgggaa 13980
cttgactgga tcacgaatga agaagaatgg gaaattcaac atgacccaat cccaattacc 14040
ttcagtagtg tagaacttac aagccatacc tttcaaatct ctcatacctt cgttagaacc 14100
tctttccaaa ccagtggtag agaatctagt aacacatgga gtctttttac cgacaccaac 14160
caacatatcg atatcgcaaa tatcggagat atcggcagta acttcaaatt caccataagc 14220
accagcacct ttagcgtgaa caactctttc tggaatcttt tctctgttga aatgggccaa 14280
ggtatcaacc aaatgatgat cagacaaagc caaagttcta ccatcagctg gtggaccttc 14340
aggcttatga aatggacaac cattagcagt agtgtaagtt ggcttagaat ctggagccat 14400
aagctttttg atagatttga ctgtgttatt ttgcgtgagg ttatgagtag aaaataataa 14460
ttgagaaagg aatatgacaa gaaatatgaa aataaaggga acaaacccaa atctgattgc 14520
aaggagagtg aaagagcctt gtttatatat ttttttttcc tatgttcaac gaggacagct 14580
aggtttatgc aaaaatgtgc catcaccata agctgattca aatgagctaa aaaaaaaata 14640
gttagaaaat aaggtggtgt tgaacgatag caagtagatc aagacaccgt ctaacagaaa 14700
aaggggcagc ggacaatatt atgcaattat gaagaaaagt actcaaaggg tcggaaaaat 14760
attcaaacga tatttgcata aaatcctcaa ttgattgatt attccatagt aaaataccgt 14820
aacaacacaa aattgttctc aaattcataa attattcatt ttttccacga gcctcatcac 14880
acgaaaagtc agaagagcat acataatctt ttaaatgcat aggttatgca ttttgcaaat 14940
gccaccaggc aacaaaaata tgcgtttagc gggcggaatc gggaaggaag ccggaaccac 15000
caaaaactgg aagctacgtt tttaaggaag gtatgggtgc agtgtgctta tctcaagaaa 15060
tattagttat gatataaggt gttgaagttt agagataggt aaataaacgc ggggtgtgtt 15120
tattacatga agaagaagtt agtttctgcc ttgcttgttt atcttgcaca tcacatcagc 15180
ggaacatatg ctcacccagt cgcatggcgc gt 15212
<210> 174
<211> 13250
<212> DNA
<213> Artificial Sequence
<220>
<223> HRT5 containing EasD, EasH, EasA, and EasG
<400> 174
accacggtga acaatccccg ctggctcata tttgccgccg gttcccgtaa atcctccggt 60
acgctccagc gtgttttatc tctgcgagca taatgcctgc gtcatccgcc agcaggagct 120
ggactttact gatgcccgtt atatctgcga aaagaccggg atctggaccc gtgatggcat 180
tctctggttt tcgtcatccg gtgaagagat tgagccacct gacagtgtga cctttcacat 240
ctggacagcg tacagcccgt tcaccacctg ggtgcagatt gtcaaagact ggatgaaaac 300
gaaaggggat acgggaaaac gtaaaacctt cgtaaacacc acgctcggtg agacgtggga 360
ggcgaaaatt ggcgaacgtc cggatgctga agtgatggca gagcggaaag agcattattc 420
agcgcccgtt cctgaccgtg tggcttacct gaccgccggt atcgactccc agctggaccg 480
ctacgaaatg cgcgtatggg gatgggggcc gggtgaggaa agctggctga ttgaccggca 540
gattattatg ggccgccacg acgatgaaca gacgctgctg cgtgtggatg aggccatcaa 600
taaaacctat acccgccgga atggtgcaga aatgtcgata tcccgtatct gctgggatac 660
tggcgttttc ccgtctttca gtgccttgtt cagttcttcc tgacgggcgg tatatttctc 720
cagcttacgc gccgttaact gtcagaccaa gtttactcat atatacttta gattgattta 780
aaacttcatt tttaatttaa aaggatctag gtgaagatcc tttttgataa tctcatgacc 840
aaaatccctt aacgtgagtt ttcgttccac tgagcgtcag accccgtaga aaagatcaaa 900
ggatcttctt gagatccttt ttttctgcgc gtaatctgct gcttgcaaac aaaaaaacca 960
ccgctaccag cggtggtttg tttgccggat caagagctac caactctttt tccgaaggta 1020
actggcttca gcagagcgca gataccaaat actgttcttc tagtgtagcc gtagttaggc 1080
caccacttca agaactctgt agcaccgcct acatacctcg ctctgctaat cctgttacca 1140
gtggctgctg ccagtggcga taagtcgtgt cttaccgggt tggactcaag acgatagtta 1200
ccggataagg cgcagcggtc gggctgaacg gggggttcgt gcacacagcc cagcttggag 1260
cgaacgacct acaccgaact gagataccta cagcgtgagc tatgagaaag cgccacgctt 1320
cccgaaggga gaaaggcgga caggtatccg gtaagcggca gggtcggaac aggagagcgc 1380
acgagggagc ttccaggggg aaacgcctgg tatctttata gtcctgtcgg gtttcgccac 1440
ctctgacttg agcgtcgatt tttgtgatgc tcgtcagggg ggcggagcct atggaaaaac 1500
gccagcaacg cggccttttt acggttcctg gccttttgct ggccttttgc tcacatgttc 1560
tttcctgcgt tatcccctga ttctgtggat aaccgtatta ccgcctttga gtgagctgat 1620
accgctcgcc gcagccgaac gaccgagcgc agcgagtcag tgagcgagga agcggaagag 1680
cgcccaatac gcatgccacg ttcgctcgcg tatcggtgat tcattctgct aaccagtaag 1740
gcaaccccgc cagcctagcc gggtcctcaa cgacaggagc acgatcatgc gcacccgtgg 1800
ccaggaccca acgctgcggg gggggggggg gggttttctt tccaattttt tttttttcgt 1860
cattataaaa atcattacga ccgagattcc cggccgggta ataactgata taattaaatt 1920
gaagctctaa tttgtgagtt tagtatacat gcatttactt ataatacagt tttttagttt 1980
tgctggccgc atcttctcaa atatgcttcc cagcctgctt ttctgtaacg ttcaccctct 2040
accttagcat cccttccctt tgcaaatagt cctcttccaa caataataat gtcagatcct 2100
gtagagacca catcatccac ggttctatac tgttgaccca atgcgtctcc cttgtcatct 2160
aaacccacac cgggtgtcat aatcaaccaa tcgtaacctt catctcttcc acccatgtct 2220
ctttgagcaa taaagccgat aacaaaatct ttgtcgctct tcgcaatgtc aacagtaccc 2280
ttagtatatt ctccagtaga tagggagccc ttgcatgaca attctgctaa catcaaaagg 2340
cctctaggtt cctttgttac ttcttctgcc gcctgcttca aaccgctaac aatacctggg 2400
cccaccacac cgtgtgcatt cgtaatgtct gcccattctg ctattctgta tacacccgca 2460
gagtactgca atttgactgt attaccaatg tcagcaaatt ttctgtcttc gaagagtaaa 2520
aaattgtact tggcggataa tgcctttagc ggcttaactg tgccctccat ggaaaaatca 2580
gtcaagatat ccacatgtgt ttttagtaaa caaattttgg gacctaatgc ttcaactaac 2640
tccagtaatt ccttggtggt acgaacatcc aatgaagcac acaagtttgt ttgcttttcg 2700
tgcatgatat taaatagctt ggcagcaaca ggactaggat gagtagcagc acgttcctta 2760
tatgtagctt tcgacatgat ttatcttcgt ttcctgcagg tttttgttct gtgcagttgg 2820
gttaagaata ctgggcaatt tcatgtttct tcaacactac atatgcgtat atataccaat 2880
ctaagtctgt gctccttcct tcgttcttcc ttctgttcgg agattaccga atcaaaaaaa 2940
tttcaaggaa accgaaatca aaaaaaagaa taaaaaaaaa atgatgaatt gaaacccccc 3000
cccccccccc cgatgcgccg cgtgcggctg ctggagatgg cggacgcgat ggatatgttc 3060
tgccaagggt tggtttgcgc attcacagtt ctccgcaaga attgattggc tggatctgat 3120
atccctgcat ggcgcgttca tcgtccacct ccggagaaca ggccaccatc acgcatctgt 3180
gtctgaattt catcacgacg cgccttaagg gcaccaataa ctgccttaaa aaaattacgc 3240
cccgccctgc cactcatcgc agtactgttg taattcatta agcattctgc cgacatggaa 3300
gccatcacag acggcatgat gaacctgaat cgccagcggc atcagcacct tgtcgccttg 3360
cgtataatat ttgcccatgg tgaaaacggg ggcgaagaag ttgtccatat tggccacgtt 3420
taaatcaaaa ctggtgaaac tcacccaggg attggctgag acgaaaaaca tattctcaat 3480
aaacccttta gggaaatagg ccaggttttc accgtaacac gccacatctt gcgaatatat 3540
gtgtagaaac tgccggaaat cgtcgtggta ttcactccag agcgatgaaa acgtttcagt 3600
ttgctcatgg aaaacggtgt aacaagggtg aacactatcc catatcacca gctcaccgtc 3660
tttcattgcc atacggaatt ccggatgagc attcatcagg cgggcaagaa tgtgaataaa 3720
ggccggataa aacttgtgct tatttttctt tacggtcttt aaaaaggccg taatatccag 3780
ctgaacggtc tggttatagg tacattgagc aactgactga aatgcctcaa aatgttcttt 3840
acgatgccat tgggatatat caacggtggt atatccagtg atttttttct ccattttagc 3900
ttccttagct cctgaaaatc tcgataactc aaaaaatacg cccggtagtg atcttatttc 3960
attatggtga aagttggaac ctcttacgtg ccgatcaacg tctcattttc gccaaaagtt 4020
ggcccagggc ttcccggtat caacagggac accaggattt atttattctg cgaagtgatc 4080
ttccgtcaca ggtattggac caccctgtgg gtttataagc gcgctgctgg cgtgtaaggc 4140
ggtgacggcg aaggaagggt ccttttcatc acgtgctata aaaataatta taatttaaat 4200
tttttaatat aaatatataa attaaaaata gaaagtaaaa aaagaaatta aagaaaaaat 4260
agtttttgtt ttccgaagat gtaaaagact ctagggggat cgccaacaaa tactaccttt 4320
tatcttgctc ttcctgctct caggtattaa tgccgaattg tttcatcttg tctgtgtaga 4380
agaccacaca cgaaaatcct gtgattttac attttactta tcgttaatcg aatgtatatc 4440
tatttaatct gcttttcttg tctaataaat atatatgtaa agtacgcttt ttgttgaaat 4500
tttttaaacc tttgtttatt tttttttctt cattccgtaa ctcttctacc ttctttattt 4560
actttctaaa atccaaatac aaaacataaa aataaataaa cacagagtaa attcccaaat 4620
tattccatca ttaaaagata cgaggcgcgt gtaagttaca ggcaagcgat ccgtcctaag 4680
aaaccattat tatcatgaca ttaacctata aaaataggcg tatcacgagg ccctttcgtc 4740
tcgcgcgttt cggtgatgac ggtgaaaacc tctgacacat gcagctcccg gagacggtca 4800
cagcttgtct gtaagcggat gccgggagca gacaagcccg tcagggcgcg tcagcgggtg 4860
ttggcgggtg tcggggctgg cttaactatg cggcatcaga gcagattgta ctgagagtgc 4920
accacggcgc gtggcaccct tgcgggccat gtcatacacc gccttcagag cagccggacc 4980
tatctgcccg ttacgcgcca gcttgcaaat taaagccttc gagcgtccca aaaccttctc 5040
aagcaaggtt ttcagtataa tgttacatgc gtacacgcgt ctgtacagaa aaaaaagaaa 5100
aatttgaaat ataaataacg ttcttaatac taacataact ataaaaaaat aaatagggac 5160
ctagacttca ggttgtctaa ctccttcctt ttcggttaga gcggatgtgg ggggagggcg 5220
tgaatgtaag cgtgacataa ctaattacat gatatcgaca aaggaaaagg gggacggatc 5280
tccgaggcct cggacccgtc gggccgccgt cggacgtgcc gcggttatgg agttgcagca 5340
ccaacgttaa tattggcacc gtaaactggt ctagaatctt cagacaacaa ccagacgata 5400
gttctagcaa catcttcagg ttgaatgatt gggaaacctt ctttgatgta ggcttctttt 5460
acttcgtcga tagatttagc ttgtggcatg aattgtggca aagctggagt atcggtaata 5520
cctggagaaa cagaattgat tctaataccc aatggccaag catccatagc agcacaagaa 5580
gtaaagtaag cacaagcacc tttagaagta ccgtaagcat aagcatctgg caaatggata 5640
aaggcagcaa tggaaccaat gttaacgata gctctatctt ttggacctgg caaagccttc 5700
atagctctaa taccagctct agtacagtag aaaacaccgt ccaagttaac cttgaaaact 5760
ctttgccatt cttcatcagt ttcttccaac aaagctggag cttgtctagc accagctggt 5820
tgagcaatac cagcaacatt agccaaacca tgcaagtcac caaaagtttg aacgatagtt 5880
tgcaaccaag catcaacttc agcggatgaa gaaacatcca atctagtgca atgaaccttt 5940
gtagctggat tcaaggcttg aatatcagag gtaacggatt cgaaaccctt tggagaaata 6000
tcaccaatgc atataacagc agcacctcta gcagccaata atctacaagt agcagcacca 6060
ataccagaag caccaccagt aacagcgaaa attctagagt tgacaacggt cataagcttt 6120
ttgtttgttt atgtgtgttt attcgaaact aagttcttgg tgttttaaaa ctaaaaaaaa 6180
gactaactat aaaagtagaa tttaagaagt ttaagaaata gatttacaga attacaatca 6240
atacctaccg tctttatata cttattagtc aagtagggga ataatttcag ggaactggtt 6300
tcaacctttt ttttcagctt tttccaaatc agagagagca gaaggtaata gaaggtgtaa 6360
gaaaatgaga tagatacatg cgtgggtcaa ttgccttgtg tcatcattta ctccaggcag 6420
gttgcatcac tccattgagg ttgtgtccgt tttttgcctg tttgtgcccc tgttctctgt 6480
agttgcgcta agagaatgga cctatgaact gatggttggt gaagaaaaca atattttggt 6540
gctgggattc tttttttttc tggatgccag cttaaaaagc gggctccatt atatttagtg 6600
gatgccagga ataaactgtt cacccagaca cctacgatgt tatatattct gtgtaacccg 6660
ccccctattt tgggcatgta cgggttacag cagaattaaa aggctaattt tttgactaaa 6720
taaagttagg aaaatcacta ctattaatta tttacgtatt ctttgaaatg gcagtattga 6780
taatgataaa ctcgaactgg gcgcgtcgtg ccgtcgttgt taatcaccac atggttattc 6840
tgctcaaacg tcccggacgc ctgcgaacgc gccgaaggaa aatgagaaat atcgagggag 6900
acgattcaga ggagcaggac aaactataac cgactgtttg ttggaggatg ccgtacataa 6960
cgaacactgc tgaagctacc atgtctacag tttagaggaa tgggtacaac tcacaggcga 7020
gggatggtgt tcactcgtgc tagcaaacgc ggtgggagca aaaagtagaa tattatcttt 7080
tattcgtgaa acttcgaaca ctgtcatcta aagatgctat atactaatat aggcatactt 7140
gataatgaaa actataaatc gtaaagacat aagagatccg cgggacaaat gtggatcttc 7200
gatgtggttt tccataaacc aagttggtct taagacgacg tacttaacac cttctttaga 7260
atccaaatga gcatgagctt cacccatcat tggatcacct ttttcagtgt tagaagcaga 7320
aaccaaagcg tatcttctaa cacctctaga atgagccaaa tcgatgaatt cgaacattgg 7380
atgaacaaat tctggagcat gagcaccaac caaatagata gcagtaatag gttctggagc 7440
ttgatcccaa gcattagtcc aagtatcctt gttcaaccag tcaaaatgag cggttctata 7500
agtagcaatt ggagcagtag aagaagtacc gacaacaaaa gggatttgag cttcatgcaa 7560
caaagcagac aatctagaag cagttttacc tctaccaccc aaaaccaaga tggtcattta 7620
caaccaatgt tctctgtttt ctctagcgta atcaacaaat cttctaggtg gtctaccagt 7680
aaccttttca acgtcttgag aaactctttc ttcagcacca gtagcaacag cagcttccat 7740
agtagccaac atttcagcaa aatctggtgg caaaccagca gtatcaacca agaattgacc 7800
gaacttttca gctggcaaag taacatgggt aatctttcta cccaacaaat cggtcaagat 7860
ttcagccata tcagcataag acaacaagtc tggacccaaa accatatatt gttgttcttt 7920
ttgggtcaat ggttgagtca aagcatgaaa agcaactctg gcaatatcat cagcagaaac 7980
aaatggaatc ttacctggac cagtagcaga gtagattttg ttttcggtct taatccaaaa 8040
gctttgtttt atatttgttg taaaaagtag ataattactt ccttgatgat ctgtaaaaaa 8100
gagaaaaaga aagcatctaa gaacttgaaa aactacgaat tagaaaagac caaatatgta 8160
tttcttgcat tgaccaattt atgcaagttt atatatatgt aaatgtaagt ttcacgaggt 8220
tctactaaac taaaccaccc ccttggttag aagaaaagag tgtgtgagaa caggctgttg 8280
ttgtcacacg attcggacaa ttctgtttga aagagagaga gtaacagtac gatcgaacga 8340
actttgctct ggagatcaca gtgggcatca tagcatgtgg tactaaaccc tttcccgcca 8400
ttccagaacc ttcgattgct tgttacaaaa cctgtgagcc gtcgctagga ccttgttgtg 8460
tgacgaaatt ggaagctgca atcaatagga agacaggaag tcgagcgtgt ctgggttttt 8520
tcagttttgt tctttttgca aacaaatcac gagcgacggt aatttctttc tcgataagag 8580
gccacgtgct ttatgagggt aacatcaatt caagaaggag ggaaacactt cctttttctg 8640
gccctgataa tagtatgagg gtgaagccaa aataaaggat tcgcgcccaa atcggcatct 8700
ttaaatgcag gtatgcgata gttcctcact ctttccttac tcacgagtaa ttcttgcaaa 8760
tgcctattat gcagatgtta taatatctgt gcgtggcgcg tccggctgtc tgccatgctg 8820
cccggtgtac cgacataacc gccggtggca tagccgcgca tacgcgccat ttccttccat 8880
cttgtgattc atgctatcca tcttttttga gtatccaatt aacgaagacg ttaccagctg 8940
attgaaggtt ctcaaagtga ctgtactcca tgttttctta tcatccatgt agttattttt 9000
caaactgcaa attcaagaaa aagccacgcg tgtgcacctt ttttttcccc ttccagtgca 9060
ttatgcaata gacagcacga gtctttgaaa aagtaactta taaaactgta tcaattttta 9120
aacctaaata gattcataaa ctattcgtta atataaagtg ttctaaacta tgatgaaaaa 9180
ataagcagaa aagactaata attcttagtt aaaagcactc cgcggttagg ccttcttagc 9240
aacttgatta gacttcaagt caataccggt agccaaatcc ttcaaatcgt aggtccaaat 9300
atcaacaccc ttagcagaaa cagttctcca accaaccatt ttttgggcca atggagtcat 9360
agtttcaacc aattctcttg gcaaagcaac actagcttca actggagtga attgacaaga 9420
ttgcatgatc aatgggaaac ctcttctcaa aaaatcagta gtgttgttct taccggcacc 9480
atgtaaagtt ttaccagtaa acaaaacagc atcacctgga ttcataacag ctggaatagc 9540
agtatcagct tttggtggtg gagcagaaaa atcttcccac aaatgagaac ctggcaaaac 9600
tctagtagca ccattttcag cagtgaaatc agtcaaagca gtcaagaagt tcaacataga 9660
ttctggagat tgtctaccca aggtagtaca gattggaata ccatcatgtt ctctatgcaa 9720
ttcttgacca tggtaacctg gttcagtttc caaaacagaa gaagtagtca accagtaatc 9780
accagattct ggaccgaata atctcttgca caaaccgtgc aataattcgt cgttcatcaa 9840
ttcatctctg aacaacttgg agaacaaagg caaagatgac aattgtctca taccagccaa 9900
gtaagattct tggtaggatt tttgacccaa ttcccattgc ttcatgtgtg gatcaatttc 9960
accgttgaat cttctaactt ggtcagcaga catgaaacct tcgataacaa caacgccatc 10020
ttcttgcaag acttggaaaa tagcttcatc accagctgat cttggaattc ttctcaaggt 10080
tggcttagaa gtttcggtaa tggtcataag ctttttgtaa ttaaaactta gattagattg 10140
ctatgctttc tttctaatga gcaagaagta aaaaaagttg taatagaaca agaaaaatga 10200
aactgaaact tgagaaattg aagaccgttt attaacttaa atatcaatgg gaggtcatcg 10260
aaagagaaaa aaatcaaaaa aaaaaatttt caagaaaaag aaacgtgata aaaattttta 10320
ttgccttttt cgacgaagaa aaagaaacga ggcggtctct tttttctttt ccaaaccttt 10380
agtacgggta attaacgaca ccctagagga agaaagaggg gaaatttagt atgctgtgct 10440
tgggtgtttt gaagtggtac ggcgatgcgc ggagtccgag aaaatctgga agagtaaaaa 10500
aggagtagaa acattttgaa gctaggcgcg tcagccggta aagattcccc acgccaatcc 10560
ggctggttgc ctccttcgtg aagacaaact cacgcgcctc caaaatgagc tatcaaaaac 10620
gatagatcga ttaggatgac tttgaaatga ctccgcagtg gactggccgt taatttcaag 10680
cgtgagtaaa atagtgcatg acaaaagatg agctaggctt ttgtaaaaat atcttacgtt 10740
gtaaaatttt agaaatcatt atttccttca tatcattttg tcattgacct tcagaagaaa 10800
agagccgacc aataatataa ataaataaat aaaaataata ttccattatt tctaaacaga 10860
ttcaatactc attaaaaaac tatatcaatt aatttgaatt aaccgcggtc acaatctagc 10920
tggaataaaa gattcttcag ctggagcttc agacttttgc aacaaggttt caacaccttt 10980
ttgaattcta gcagccaaag ctggagaaac caacttccag gtttcgtaaa cttgcttttg 11040
aatttcggct ctggtttgag caacagattc aaccatgttt tggaccatgg tatcttggaa 11100
ttcttgacct ctaaaatctg gcaaaacttc gtagaaatct ctggcgaact tgtaatcatg 11160
ttcagtagtt tgcaaccagg acttggattg gatttctttc aaccattcat tatactttgg 11220
atctggttct ctaggttcgg caaatctaac tggttgttgt tgagtcaaga aacctggatg 11280
agcaccataa ttagcatcaa aagtaccttg accatctcta gcaattgggt taaaggtatg 11340
agcggatcta ttaactggga tttgttgatg gttagcaccc aatctatatc tttgagcgtc 11400
tgggtaagcg aaaattctag cttgtaaaat tggatcttca gatggagcaa cacctggaac 11460
catgttagat ggagagtaag ccaattgttc aatttcggcg aagtagttag ctgggtttct 11520
tttcaaagtc aacttaccga aaacctttgg tgggatcaaa gtaacatctt ttgggtaagt 11580
acccaagttc cagtgcttag taacatccaa aatgttgtaa cccaagtttg gagcatcttt 11640
tggatcaaca acttgaacag aagcagtcca agttgggtat tcacctcttt caatagcttc 11700
atacaagtct ctagtagcgt gatctggatc caaatcattt gtacctttag cttgttgacc 11760
atcggtgaaa tttggacctc tatcagatga caagaagatg tggacgtact taaaagaacc 11820
atctggctta acccatttgt aagcatgagc catataacca gacatggatc tccaattgaa 11880
catagtaccg aaatcagaaa attgcaacaa gaccatgtgc aaggattcat gattgttggt 11940
aacccaatcc cagtacatgt ttgggttcaa caaattagtt tgtggatctc ttctttgagc 12000
gtgcatcaaa gatgggaact tgactggatc acgaatgaag aagaatggga aattcaacat 12060
gacccaatcc caattacctt cagtagtgta gaacttacaa gccatacctt tcaaatctct 12120
cataccttcg ttagaacctc tttccaaacc agtggtagag aatctagtaa cacatggagt 12180
ctttttaccg acaccaacca acatatcgat atcgcaaata tcggagatat cggcagtaac 12240
ttcaaattca ccataagcac cagcaccttt agcgtgaaca actctttctg gaatcttttc 12300
tctgttgaaa tgggccaagg tatcaaccaa atgatgatca gacaaagcca aagttctacc 12360
atcagctggt ggaccttcag gcttatgaaa tggacaacca ttagcagtag tgtaagttgg 12420
cttagaatct ggagccataa gctttttgat agatttgact gtgttatttt gcgtgaggtt 12480
atgagtagaa aataataatt gagaaaggaa tatgacaaga aatatgaaaa taaagggaac 12540
aaacccaaat ctgattgcaa ggagagtgaa agagccttgt ttatatattt ttttttccta 12600
tgttcaacga ggacagctag gtttatgcaa aaatgtgcca tcaccataag ctgattcaaa 12660
tgagctaaaa aaaaaatagt tagaaaataa ggtggtgttg aacgatagca agtagatcaa 12720
gacaccgtct aacagaaaaa ggggcagcgg acaatattat gcaattatga agaaaagtac 12780
tcaaagggtc ggaaaaatat tcaaacgata tttgcataaa atcctcaatt gattgattat 12840
tccatagtaa aataccgtaa caacacaaaa ttgttctcaa attcataaat tattcatttt 12900
ttccacgagc ctcatcacac gaaaagtcag aagagcatac ataatctttt aaatgcatag 12960
gttatgcatt ttgcaaatgc caccaggcaa caaaaatatg cgtttagcgg gcggaatcgg 13020
gaaggaagcc ggaaccacca aaaactggaa gctacgtttt taaggaaggt atgggtgcag 13080
tgtgcttatc tcaagaaata ttagttatga tataaggtgt tgaagtttag agataggtaa 13140
ataaacgcgg ggtgtgttta ttacatgaag aagaagttag tttctgcctt gcttgtttat 13200
cttgcacatc acatcagcgg aacatatgct cacccagtcg catggcgcgt 13250
<210> 175
<211> 607
<212> PRT
<213> Artificial Sequence
<220>
<223> Arthoderma otae sequence with improved N-terminus
<400> 175
Met Gln Ser Leu Phe Leu Thr Thr Gly Leu Val Ala Leu Leu Ser Trp
1 5 10 15
Leu Val Tyr Thr Gly Arg Asn Gln Val Thr Ser Cys Arg Cys Arg Pro
20 25 30
Trp Gln Pro Cys Trp Pro Ser Glu Glu Leu Trp Asn Ser Phe Asn Thr
35 40 45
Ser Ile Asp Gly Lys Leu Gln Gln Leu Lys Pro Ala Ala His Val Cys
50 55 60
Tyr Glu Pro Asn Phe Asp Lys Gly Ala Cys Asp Asp Leu Leu Arg Leu
65 70 75 80
Ser Arg Asp Ser Gly Trp Arg Ala Ser His Pro Gly Val Leu Gln Asp
85 90 95
Trp Val Trp Glu Ala Gly Glu Ser Ala Asn Glu Thr Cys Pro Met Gly
100 105 110
Ser Leu Gln Thr Ala Thr Ala Ala Lys Ser Cys His Gln Gly Arg Ile
115 120 125
Pro Leu Tyr Ser Ala Thr Val Glu Ser Ala Gln Gln Val Gln Gln Ala
130 135 140
Val Arg Phe Ala Arg Arg His Asn Leu Arg Leu Val Ile Arg Asn Thr
145 150 155 160
Gly His Asp Leu Ala Gly Arg Ser Ser Ala Pro Asp Ser Phe Gln Ile
165 170 175
His Thr His Arg Leu Gln Glu Thr Gln Phe His Thr Asp Leu Arg Leu
180 185 190
Asn Gly Ser Thr Ala Ser Leu Gly Pro Ala Val Thr Val Gly Ala Gly
195 200 205
Val Met Met Gly Asn Leu Tyr Ala Arg Ala Ala Arg Glu Gly Tyr Met
210 215 220
Val Leu Gly Gly Asp Cys Pro Thr Val Gly Val Ala Gly Gly Phe Leu
225 230 235 240
Gln Gly Gly Gly Val Ser Asp Phe Leu Ser Leu Asn Gln Gly Leu Gly
245 250 255
Val Asp Asn Val Leu Glu Tyr Glu Ile Val Thr Ala Asp Gly Glu Leu
260 265 270
Leu Val Ala Asn Thr Leu Gln Asn Gln Glu Leu Phe Trp Ala Leu Arg
275 280 285
Gly Gly Gly Gly Gly Thr Phe Gly Val Val Thr Arg Ala Thr Met Arg
290 295 300
Val Phe Pro Asp Val Pro Ala Val Ile Ser Glu Val Leu Leu Gln Ala
305 310 315 320
Pro Gln Thr Asn Ser Ser Ser Trp Thr Glu Gly Leu Ser Val Ile Leu
325 330 335
Asn Ala Leu Gln Ser Leu Asn Arg Asp Asp Val Gly Gly Gln Leu Val
340 345 350
Ile Ala Val Gln Pro Glu Leu Ala Val Gln Ala Ser Ile Lys Phe Phe
355 360 365
Phe Leu Asn Ser Thr Glu Thr Thr Ile Ile Asp Glu Arg Met Lys Ser
370 375 380
Leu Leu Thr Asp Leu Asn Arg Ile Asp Ile Gln Tyr Thr Leu Ser Ser
385 390 395 400
Lys Ala Leu Pro His Phe Ser Ser Asn Tyr Arg Gln Val Pro Asp Ile
405 410 415
His Ser Asp Asn Asp Tyr Gly Val Ile Gly Ser Thr Val Ala Ile Ser
420 425 430
Lys Glu Leu Phe Asp Ser Ser Gln Gly Pro Gln Lys Ile Ala Arg Ala
435 440 445
Leu Ala Asn Leu Pro Met Ser Pro Gly Asp Leu Leu Phe Thr Ser Asn
450 455 460
Leu Gly Gly Arg Val Ile Ser Asn Gly Glu Ile Ala Glu Thr Ser Met
465 470 475 480
His Pro Ala Trp Arg Ala Ala Ser Gln Leu Leu Asn Tyr Ile His Ala
485 490 495
Val Gly Pro Ser Ile Glu Ser Arg Val Asn Ala Leu Glu Arg Leu Thr
500 505 510
Asn Val Gln Met Pro Met Leu Tyr Ala Ile Asp Pro Asn Phe Arg Leu
515 520 525
Ser Tyr Arg Asn Val Gly Asp Pro Asn Glu Lys Asp Phe Gln Gln Val
530 535 540
Tyr Trp Gly Ser Asn Tyr Lys Arg Leu Ser Gln Ile Lys Lys Arg Trp
545 550 555 560
Asp Ser Asp Gly Leu Phe Phe Ser Lys Leu Gly Val Gly Ser Glu Leu
565 570 575
Trp Asp Ser Glu Gly Met Cys Arg Lys Asn Gln Ser Val Val Arg Gln
580 585 590
Ala Val Asn Tyr Leu Met Ser Phe Ala Thr Ser Met Val Glu Gly
595 600 605
<210> 176
<211> 602
<212> PRT
<213> Artificial Sequence
<220>
<223> Aspergillus fumigatus sequence with improved N-terminus
<400> 176
Met Pro Phe Val Phe Ser Phe Leu Leu Gly Ala Phe Leu Ser Cys Ile
1 5 10 15
Trp Leu Ser Leu Ser Gly Gly Ser Thr Cys Arg Cys Arg Pro Gly Glu
20 25 30
Ser Cys Trp Pro Ser Pro Ser Glu Trp Ala Ala Leu Asn Thr Thr Leu
35 40 45
Arg Gly Asp Leu Val Glu Val Arg Pro Ile Ala His Ile Cys His Asp
50 55 60
Pro Phe Tyr Asp His Ser Ala Cys Gln Asn Leu Arg Asp Leu Ala Lys
65 70 75 80
Asp Ser Gly Trp Arg Ala Ser Gln Ala Gly Thr Leu Gln Gly Trp Val
85 90 95
Trp Glu Val Gly Arg Thr Glu Asp Glu Thr Cys His Ala His Ser Pro
100 105 110
Arg Gly Ala Pro Cys His Gln Gly Arg Ile Pro Leu Tyr Ser Ala Ala
115 120 125
Val Glu Ser Val Asp Gln Ile Gln Val Ala Val Arg Phe Ala Gln Arg
130 135 140
His Arg Leu Arg Leu Val Val Arg Asn Thr Gly His Asp Thr Ala Gly
145 150 155 160
Arg Ser Ser Gly Ser Asp Ser Phe Gln Ile His Cys His Arg Met Lys
165 170 175
Gln Ile Glu Tyr His Asp Asn Phe Arg Ala Leu Gly Ser Asp Ile Asp
180 185 190
Arg Gly Pro Ala Val Ser Val Gly Ala Gly Val Thr Leu Gly Glu Met
195 200 205
Tyr Ala Arg Gly Ala Arg Asp Gly Trp Val Val Val Gly Gly Glu Cys
210 215 220
Pro Thr Val Gly Ala Ala Gly Gly Phe Leu Gln Gly Gly Gly Val Ser
225 230 235 240
Ser Phe His Ser Phe Ile Asp Gly Leu Ala Val Asp Asn Val Leu Glu
245 250 255
Phe Glu Val Val Thr Ala Lys Gly Asp Val Val Val Ala Asn Asp His
260 265 270
Gln Asn Pro Asp Ile Phe Trp Ala Leu Arg Gly Gly Gly Gly Gly Thr
275 280 285
Phe Gly Ile Val Thr Arg Ala Thr Met Arg Val His Leu Asn Ser Pro
290 295 300
Val Cys Val Ser Glu Val Ala Val Ser Gly Leu Arg Asn Asn Ser Leu
305 310 315 320
Leu Trp Thr Lys Gly Ile Thr Gly Leu Phe Ser Ile Leu Arg Ser Phe
325 330 335
Asn Gln Gln Gly Ile Pro Gly Gln Phe Ile Leu Arg Pro Leu Ser Lys
340 345 350
Asp Gln Val Asn Ala Ser Leu Thr Leu Tyr Ser Leu Asn Thr Asp Asp
355 360 365
Thr Arg Arg Ser Ala Glu Asn Met Leu Ser Ile Arg Asn Ile Leu Glu
370 375 380
Ser Thr Thr Leu Pro Phe Thr Leu Ala Ser Arg Cys Leu Pro Lys Ile
385 390 395 400
Ser Asp Ala Leu Arg Lys Gly Pro Asp Met Leu Pro Val Asn Tyr Gly
405 410 415
Ile Ile Thr Gly Ser Val Leu Val Ser Glu Asp Leu Phe Asn Ser Glu
420 425 430
Glu Gly Pro Leu His Leu Ala Lys Gln Leu Glu His Phe Pro Met Gly
435 440 445
Pro Met Asp Leu Leu Phe Thr Ser Asn Leu Gly Gly Asn Val Ser Ala
450 455 460
Asn Thr Gly Lys Lys His Arg Asp Thr Ser Met His Pro Gly Trp Arg
465 470 475 480
Gln Ala Ala His Leu Ile Asn Phe Val Arg Ser Val Ser Thr Pro Thr
485 490 495
Ala His Glu Lys Ala Arg Ser Leu Glu Glu Leu His Ser Val Gln Met
500 505 510
Arg Gln Leu Tyr Asp Ile Glu Pro Asp Phe Arg Val Ser Tyr Arg Asn
515 520 525
Leu Gly Asp Pro Leu Glu Ser Asp Ala Ala Gln Val Tyr Trp Gly Pro
530 535 540
Asn Tyr Lys Arg Leu Leu Glu Ile Lys Arg Lys Trp Asp Pro Glu Asp
545 550 555 560
Leu Phe Phe Ser Gln Leu Gly Val Gly Ser Glu Gly Trp Thr Glu Asp
565 570 575
Gln Met Cys Lys Arg Gln Gln Arg Leu Gln Gln Met Leu Gln Tyr Leu
580 585 590
Met Ser Ser Ile Ala Gln Arg Val Tyr Arg
595 600
<210> 177
<211> 607
<212> PRT
<213> Artificial Sequence
<220>
<223> Arthroderma gypseum sequence with improved N-terminus
<400> 177
Met Gln Phe Leu Phe Leu Thr Thr Gly Leu Val Ala Leu Leu Ser Trp
1 5 10 15
Leu Ile Tyr Thr Gln Glu Thr Gln Pro Ala Ser Cys Arg Cys Arg Pro
20 25 30
Trp Glu Ser Cys Trp Pro Ser Glu Glu Ile Trp Asn Ser Leu Asn Ser
35 40 45
Ser Val Asp Gly Asn Leu His Arg Leu Lys Pro Ala Ala His Val Cys
50 55 60
Tyr Gly Ser Ser Phe Asn Gln Ser Ala Cys Asp Asp Met Leu Leu Leu
65 70 75 80
Ser Arg Asn Ser Gly Trp Arg Ala Ser Asn Pro Gly Val Leu Gln Asp
85 90 95
Trp Val Trp Glu Ala Gly Glu Thr Ala Asn Glu Thr Cys Pro Val Gly
100 105 110
Ser Leu Arg Thr Val Ser Ala Val Asn Ser Cys His Gln Gly Arg Ile
115 120 125
Pro Leu Tyr Ser Val Gly Val Glu Ser Ala Gln Gln Val Gln Lys Ala
130 135 140
Val Arg Phe Ala Arg Arg His Asn Leu Arg Leu Val Ile Arg Asn Thr
145 150 155 160
Gly His Asp Leu Ala Gly Arg Ser Ser Ala Pro Asp Ser Phe Gln Ile
165 170 175
His Thr His Arg Leu Gln Glu Ile His Phe His Thr Asn Leu Arg Leu
180 185 190
Asn Gly Ser Asn Ala Ser Leu Gly Pro Ala Val Thr Val Gly Ala Gly
195 200 205
Val Met Met Gly Asn Leu Tyr Ser Gln Ala Ala Arg Asn Gly Tyr Met
210 215 220
Val Leu Gly Gly Asp Cys Pro Thr Val Gly Val Val Gly Gly Phe Leu
225 230 235 240
Gln Gly Gly Gly Val Ser Asp Phe Leu Ser Leu Asn Gln Gly Leu Gly
245 250 255
Val Asp Asn Val Leu Glu Tyr Glu Val Val Thr Ala Asp Gly Glu Ile
260 265 270
Val Val Ala Asn Ala Leu Gln Asn Gln Asp Leu Phe Trp Ala Leu Arg
275 280 285
Gly Gly Gly Gly Gly Thr Phe Gly Val Val Thr Arg Ala Thr Met Arg
290 295 300
Val Phe Pro Asp Val Pro Val Val Ile Ser Glu Ile Leu Leu Glu Ala
305 310 315 320
Ser Lys Ser Asn Ser Ser Ser Trp Thr Gln Gly Leu Ser Val Val Leu
325 330 335
Thr Ala Leu Gln Ser Leu Asn Arg Glu Asn Val Gly Gly Gln Leu Val
340 345 350
Ile Val Val Leu Pro Lys Leu Ala Val Gln Ala Ser Ile Lys Phe Phe
355 360 365
Phe Leu Asp Ala Thr Glu Thr Ala Ala Ile Asp His Arg Met Lys Ser
370 375 380
Phe Leu Thr Glu Leu Ser Arg Ala Asn Val Lys Tyr Ala Tyr Ser Ser
385 390 395 400
Lys Gly Leu Pro His Phe Ser Ser Asn Tyr Arg Gln Val Pro Asp Ile
405 410 415
His Ala Asp Asn Asp Tyr Gly Val Leu Gly Ser Thr Val Ala Ile Ser
420 425 430
Gln Gln Leu Phe Asp Ser Pro Gln Gly Pro Glu Lys Val Ala Asn Ala
435 440 445
Leu Ala Asn Leu Pro Met Ser Pro Gly Asp Leu Leu Phe Thr Ser Asn
450 455 460
Leu Gly Gly Arg Val Ile Leu Asn Gly Gly Leu Ala Glu Thr Ser Met
465 470 475 480
His Pro Ala Trp Arg Ala Ala Ser Gln Leu Leu Asn Tyr Val His Thr
485 490 495
Val Glu Pro Ser Ile Glu Gly Arg Ala Lys Ala Arg Glu Lys Leu Thr
500 505 510
Asn Ile Gln Met Pro Met Leu Tyr Ala Ile Asp Pro Lys Phe Lys Leu
515 520 525
Ser Tyr Arg Asn Val Gly Asp Pro Asn Glu Lys Asn Phe Gln Gln Val
530 535 540
Tyr Trp Gly Ser Asn Tyr Gly Arg Leu Ser Asp Ile Lys Lys Lys Trp
545 550 555 560
Asp Thr Thr Asp Leu Phe Phe Ser Lys Leu Gly Ile Gly Ser Glu Lys
565 570 575
Trp Asp Ser Glu Gly Met Cys Arg Lys His Gln Glu Ala Phe Tyr Gln
580 585 590
Lys Met Arg Arg Phe Ile Ser Leu Thr Thr Phe Thr Leu Lys Asn
595 600 605
<210> 178
<211> 583
<212> PRT
<213> Artificial Sequence
<220>
<223> Arthroderma benhamiae sequence with improved N-terminus
<400> 178
Met Gln Phe Leu Leu Trp Ser Thr Gly Leu Val Ala Leu Leu Ser Trp
1 5 10 15
Leu Ile Tyr Thr Gln Glu Thr Gln Pro Ala Ser Cys Arg Cys Arg Pro
20 25 30
Trp Glu Ser Cys Trp Pro Ser Glu Glu Leu Trp Asn Ser Phe Asn Thr
35 40 45
Ser Val Asp Gly Lys Leu His Arg Leu Arg Pro Ala Ala His Val Cys
50 55 60
Tyr Gly Pro Ser Phe Asn Arg Ser Ala Cys Asp Asn Ile Leu Leu Leu
65 70 75 80
Ser Arg Asp Ser Gly Trp Arg Ala Ser Asn Pro Gly Val Leu Gln Asp
85 90 95
Trp Val Trp Glu Ala Gly Glu Thr Ala Asn Glu Ser Cys Pro Val Gly
100 105 110
Ser Leu Arg Thr Ala Ser Ala Val Asn Ser Cys His Gln Gly Arg Ile
115 120 125
Pro Leu Phe Thr Val Gly Val Glu Ser Thr Lys Gln Val Gln Glu Ala
130 135 140
Val Arg Phe Ala Arg Lys His Asn Leu Arg Leu Val Ile Arg Asn Thr
145 150 155 160
Gly His Asp Leu Ala Gly Arg Ser Ser Ala Pro Asp Ser Phe Gln Ile
165 170 175
His Thr His His Leu Gln Glu Ile Gln Phe His Ala Asp Met Arg Leu
180 185 190
Asp Gly Ser Asn Thr Ser Leu Gly Pro Ala Val Thr Val Gly Ala Gly
195 200 205
Val Met Met Gly Asn Leu Tyr Ala Gln Ala Ala Arg His Gly Tyr Met
210 215 220
Val Leu Gly Gly Asp Cys Pro Thr Val Gly Val Val Gly Gly Phe Leu
225 230 235 240
Gln Gly Gly Gly Ile Ser Asp Phe Leu Ser Leu Asn Gln Gly Phe Gly
245 250 255
Val Asp Asn Val Leu Glu Tyr Glu Val Val Thr Ala Asp Gly Glu Leu
260 265 270
Val Val Ala Asn Ala Leu Gln Asn Gln Asp Leu Phe Trp Ala Leu Arg
275 280 285
Gly Gly Gly Gly Gly Thr Phe Gly Val Val Thr Arg Ala Thr Met Arg
290 295 300
Val Phe Pro Asp Val Pro Val Val Ile Ser Glu Ile Leu Leu Glu Ala
305 310 315 320
Pro Gln Ala Ile Ser Ser Ser Trp Thr Gln Gly Leu Ser Ile Val Leu
325 330 335
Thr Ala Leu Gln Ser Leu Asn Arg Asp Asn Val Gly Gly Gln Leu Val
340 345 350
Ile Ala Val Leu Pro Lys Leu Ala Val Gln Ala Ser Ile Lys Phe Phe
355 360 365
Phe Leu Asp Ala Thr Glu Ala Thr Val Ile Asp Arg Arg Met Lys Pro
370 375 380
Phe Leu Thr Lys Leu Ser Arg Ala Asn Val Lys Tyr Thr Tyr Ser Ser
385 390 395 400
Lys Asn Leu Pro His Phe Ser Ser Asn Tyr Arg Gln Val Pro Asp Ile
405 410 415
His Ser Asp Asn Asp Tyr Gly Val Leu Gly Ser Thr Val Ala Ile Ser
420 425 430
Gln Gln Leu Phe Asp Ser Pro Gln Gly Pro Glu Lys Val Ala Thr Ala
435 440 445
Leu Ala Asn Leu Pro Val Ser Ala Gly Asp Leu Ile Phe Thr Ser Asn
450 455 460
Leu Gly Gly Arg Val Ile Ser Asn Gly Glu Leu Ala Glu Thr Ser Met
465 470 475 480
His Pro Ala Trp Arg Ser Ala Ser Gln Leu Ile Asn Tyr Val His Thr
485 490 495
Val Glu Pro Ser Ile Glu Gly Arg Ala Lys Ala Arg Glu Arg Leu Thr
500 505 510
Asn Thr Gln Met Pro Met Leu Tyr Ala Leu Asp Pro Asn Leu Lys Leu
515 520 525
Ser Tyr Arg Asn Val Gly Asp Pro Asn Glu Lys Asp Phe Gln Gln Ile
530 535 540
Tyr Trp Gly Pro Asn Tyr Gly Arg Leu Ser Asn Ile Lys Lys Lys Trp
545 550 555 560
Asp Thr Asp Asp Leu Phe Phe Ser Lys Leu Gly Val Gly Ser Glu Arg
565 570 575
Trp Asp Ser Glu Ala Arg Arg
580
<210> 179
<211> 429
<212> PRT
<213> Artificial Sequence
<220>
<223> DmaW variant based on Aspergillus japonicus DmaW sequence
<400> 179
Met Thr Ala Gly Gln Gly Ile Lys Thr Gly Asn Ala Ser Asp Cys Glu
1 5 10 15
Val Tyr Arg Thr Leu Ser Val Ala Leu Asp Phe Ala Asn Gln Asp Glu
20 25 30
Glu Leu Trp Trp His Ser Thr Ala Pro Met Phe Ala Gln Met Leu Gln
35 40 45
Ser Thr Asn Tyr Asn Leu His Ala Gln Tyr Lys His Leu Leu Ile Tyr
50 55 60
Lys Lys Asn Val Ile Pro Phe Leu Gly Val Tyr Pro Thr Asn Asp Lys
65 70 75 80
Pro Arg Trp Leu Ser Ile Leu Thr Arg Tyr Gly Thr Pro Phe Glu Leu
85 90 95
Ser Leu Asn Cys Ser Gly Pro Leu Val Arg Tyr Thr Tyr Glu Pro Ile
100 105 110
Asn Ala Ala Thr Gly Thr Ala Arg Asp Pro Phe Asn Thr His Ala Val
115 120 125
Trp Asp Ser Leu Glu Gln Leu Met Ala Leu Gln Ser Gly Ile Asp Leu
130 135 140
Asp Leu Phe Arg His Phe Lys Asn Asp Leu Thr Leu Ser Ala Glu Glu
145 150 155 160
Ser Glu Tyr Leu Tyr Lys Asn Asn Leu Val Gly Glu Gln Ile Arg Thr
165 170 175
Gln Asn Lys Leu Ala Leu Asp Leu Gln Asp Gly Glu Phe Val Val Lys
180 185 190
Thr Tyr Ile Tyr Pro Ala Leu Lys Ser Leu Ala Thr Gly Arg Ser Ile
195 200 205
His Glu Leu Val Phe Gly Ser Ala Phe Arg Trp Ser Lys Gln Tyr Pro
210 215 220
Glu Leu Arg Lys Pro Leu Asp Thr Leu Glu Gln Tyr Val Tyr Ser Arg
225 230 235 240
Gly Pro Ser Ser Thr Ala Ser Pro Arg Leu Leu Ser Cys Asp Leu Ile
245 250 255
Asp Pro Thr Lys Ser Arg Ile Lys Ile Tyr Leu Leu Glu Arg Met Val
260 265 270
Thr Leu Glu Ala Leu Glu Asp Leu Trp Thr Met Gly Gly Glu Arg Thr
275 280 285
Asp Ala Ser Thr Leu Ala Gly Leu Glu Met Ile Arg Glu Leu Trp Glu
290 295 300
Leu Ile Arg Leu Pro Ala Gly Leu Gln Ser Tyr Pro Ala Pro Tyr Leu
305 310 315 320
Pro Ile Gly Thr Ile Pro Asp Glu Gln Leu Pro Leu Met Ala Asn Tyr
325 330 335
Thr Ile His His Asp Asp Pro Val Pro Glu Pro Gln Val Tyr Phe Thr
340 345 350
Thr Phe Gly Arg Asn Asp Met Gln Ile Ala Asp Ala Leu Ala Thr Phe
355 360 365
Phe Glu Arg Arg Gly Trp His Glu Met Ala Arg Gln Tyr Lys Ala Glu
370 375 380
Leu Cys Ser His Tyr Pro His Ala Asp His Glu Thr Leu Asn Tyr Leu
385 390 395 400
His Ala Tyr Ile Ser Phe Ser Tyr Arg Lys Asn Lys Pro Tyr Leu Ser
405 410 415
Val Tyr Leu Gln Ser Leu Glu Thr Gly Asp Trp Val Thr
420 425
<210> 180
<211> 461
<212> PRT
<213> Paecilomyces divaricatus
<400> 180
Met Ser Ser Arg Lys Thr Arg Asn Ala Ser Ser Ser Glu Val Tyr Gln
1 5 10 15
Thr Leu Ser Ser Leu Phe Asp Phe Pro Asp Glu Glu Gln Lys Leu Trp
20 25 30
Trp His Ser Thr Ala Pro Met Phe Ala Asp Met Leu Gln Thr Ala Asn
35 40 45
Tyr Asp Thr His Ala Gln Tyr Arg His Leu Gly Ile Tyr Lys Lys Tyr
50 55 60
Val Ile Pro Phe Leu Gly Val Tyr Pro Thr Asn Asp Arg Asp Arg Trp
65 70 75 80
Leu Ser Ile Leu Thr Arg Tyr Gly Thr Pro Phe Glu Leu Ser Leu Asn
85 90 95
Cys Ser Asp Asp Val Val Arg Tyr Thr Phe Glu Pro Ile Asn Ala Ala
100 105 110
Thr Gly Thr Ile Gln Asp Pro Phe Asn Thr His Ala Ile Trp Asp Cys
115 120 125
Leu Gln Asn Phe Ser Pro Met Leu Lys Gly Val Asp Leu Glu Trp Phe
130 135 140
Ser Tyr Phe Lys Glu Lys Leu Thr Leu Asn Ala Asp Glu Ser Ala Phe
145 150 155 160
Leu Val Glu Asn Asn Leu Ala Asp Asp Pro Ile Lys Thr Gln Asn Lys
165 170 175
Leu Ala Leu Asp Leu Lys Asn Ser Gly Phe Val Leu Lys Thr Tyr Ile
180 185 190
Tyr Pro Ala Leu Lys Ser Ile Ala Thr Gly Lys Ser Ile Trp Glu Leu
195 200 205
Met Phe Asn Ser Val Ser Arg Leu Ser Pro Gln Tyr Pro Gly Leu Ala
210 215 220
Ala Pro Leu Ser Val Ile Asp Glu Tyr Val His Ser Arg Gly Pro Asn
225 230 235 240
Gly Thr Ala Gly Pro Arg Leu Phe Ser Cys Asp Leu Val His Pro Ser
245 250 255
Lys Ser Arg Ile Lys Ile Tyr Ile Leu Glu Arg Met Val Ser Leu Ala
260 265 270
Ala Met Glu Asp Leu Trp Thr Leu Gly Gly Arg Arg Lys Asp His Ser
275 280 285
Thr Leu Ala Gly Leu Asn Met Ile Arg Glu Leu Trp Glu Leu Ile Gln
290 295 300
Leu Pro Ala Gly Leu Arg Ser Tyr Pro Glu Pro Tyr Leu Pro Leu Gly
305 310 315 320
Thr Ile Pro Asn Glu Gln Leu Pro Leu Met Ala Asn Tyr Thr Leu His
325 330 335
Gln Asp Asp Pro Trp Pro Glu Pro Gln Val Tyr Phe Thr Pro Phe Gly
340 345 350
Met Asn Asp Met Ala Ile Ala Asp Ala Leu Thr Thr Phe Met Glu Arg
355 360 365
Arg Gly Trp Asn Asp Met Ala Gln Ser Tyr Lys Asp Asn Leu Cys Ser
370 375 380
Tyr Tyr Pro Asn Ala Asp His Asp His Leu Asn Tyr Val His Ala Tyr
385 390 395 400
Ile Ser Phe Ser Tyr Arg Asn Asp Lys Pro Tyr Leu Ser Val Tyr Leu
405 410 415
His Ser Phe Glu Thr Gly Asp Trp Lys Ile Ser Asn Phe Thr Asn Pro
420 425 430
Glu Ser Lys Cys Lys Glu Pro Gln Arg Val Ser Val Arg Phe Pro Val
435 440 445
Gln Gly Arg Gly Arg Gly Arg Ile Pro Ala Ser Gly Tyr
450 455 460
<210> 181
<211> 288
<212> PRT
<213> Artificial Sequence
<220>
<223> Neotyphodium lolii sequence with improved N-terminus
<400> 181
Met Thr Ile Leu Leu Thr Gly Gly Arg Gly Lys Thr Ala Ser His Ile
1 5 10 15
Ala Ser Leu Leu Gln Ala Ala Lys Val Pro Phe Ile Val Ala Ser Arg
20 25 30
Ser Ser Asp Pro Ser Ser Ser Ser Pro Tyr Tyr Gln Asn Cys Phe Asp
35 40 45
Trp Leu Asp Glu Lys Thr Tyr Gly Asp Val Leu Thr Ser Lys Asp Ser
50 55 60
Met Gln Pro Ile Ser Thr Ile Trp Leu Val Pro Pro Pro Ile Phe Asp
65 70 75 80
Leu Ala Pro Leu Met Ile Lys Phe Val Asp Phe Ala Ser Arg Lys Gly
85 90 95
Val Lys Arg Phe Val Leu Leu Ser Ala Ser Thr Ile Lys Lys Gly Gly
100 105 110
Pro Ala Met Gly Gln Val His Glu Tyr Leu Ala Ser Leu Gly Gly Ile
115 120 125
Glu Tyr Ala Val Leu Arg Pro Thr Trp Phe Met Glu Asn Phe Ser Tyr
130 135 140
Pro Gln Glu Leu Gln Arg Leu Ala Ile Lys Asn Glu Asn Lys Ile Tyr
145 150 155 160
Ser Ala Ala Gly Asp Gly Lys Leu Pro Phe Val Ser Val Ala Asp Ile
165 170 175
Ala Arg Val Ala Phe Arg Thr Leu Thr Asp Glu Lys Ser His Asn Thr
180 185 190
Asp Tyr Val Leu Leu Gly Pro Glu Leu Ile Thr Tyr Asp Gln Val Ala
195 200 205
Glu Thr Leu Ser Thr Val Leu Gly Arg Thr Ile Thr His Ile Lys Leu
210 215 220
Thr Glu Glu Glu Leu Val Lys Arg Leu Glu Asn Ser Gly Met Pro Ala
225 230 235 240
Glu Asp Ala Lys Met Leu Ala Gly Met Asp Thr Ser Ile Ser Asp Gly
245 250 255
Ala Glu Asp Arg Leu Asn Asn Val Val Lys His Val Thr Gly Ala Asp
260 265 270
Pro Arg Thr Phe Leu Asp Phe Ala Thr His Gln Lys Ala Thr Trp Gly
275 280 285
<210> 182
<211> 451
<212> PRT
<213> Artificial Sequence
<220>
<223> DmaW consensus sequence 2
<220>
<221> misc_feature
<222> (2)..(2)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (4)..(4)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (6)..(6)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (8)..(9)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (13)..(13)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (17)..(18)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (24)..(24)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (27)..(27)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (39)..(39)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (47)..(48)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (50)..(50)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (53)..(53)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (72)..(73)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (75)..(75)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (96)..(96)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (98)..(98)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (113)..(113)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (120)..(120)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (124)..(124)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (128)..(128)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (130)..(132)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (134)..(134)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (142)..(142)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (146)..(146)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (151)..(153)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (159)..(160)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (163)..(163)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (166)..(167)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (182)..(183)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (197)..(197)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (207)..(207)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (212)..(212)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (216)..(218)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (223)..(223)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (226)..(227)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (230)..(230)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (233)..(233)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (237)..(248)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (259)..(259)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (262)..(262)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (278)..(278)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (291)..(291)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (293)..(293)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (296)..(297)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (300)..(300)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (307)..(307)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (310)..(311)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (313)..(313)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (316)..(317)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (320)..(320)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (327)..(327)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (330)..(330)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (344)..(344)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (347)..(347)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (363)..(363)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (365)..(365)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (378)..(379)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (382)..(382)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (387)..(387)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (389)..(390)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (395)..(395)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (399)..(399)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (413)..(413)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (432)..(439)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (442)..(443)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (446)..(450)
<223> Xaa can be any naturally occurring amino acid
<400> 182
Met Xaa Thr Xaa Asn Xaa Ser Xaa Xaa Glu Val Tyr Xaa Thr Leu Ser
1 5 10 15
Xaa Xaa Phe Asp Phe Pro Asn Xaa Asp Gln Xaa Leu Trp Trp His Ser
20 25 30
Thr Ala Pro Met Phe Ala Xaa Met Leu Gln Thr Ala Asn Tyr Xaa Xaa
35 40 45
His Xaa Gln Tyr Xaa His Leu Gly Ile Tyr Lys Lys His Val Ile Pro
50 55 60
Phe Leu Gly Val Tyr Pro Thr Xaa Xaa Lys Xaa Arg Trp Leu Ser Ile
65 70 75 80
Leu Thr Arg Tyr Gly Thr Pro Phe Glu Leu Ser Leu Asn Cys Ser Xaa
85 90 95
Ser Xaa Val Arg Tyr Thr Tyr Glu Pro Ile Asn Ala Ala Thr Gly Thr
100 105 110
Xaa Lys Asp Pro Phe Asn Thr Xaa Ala Ile Trp Xaa Ser Leu Gln Xaa
115 120 125
Leu Xaa Xaa Xaa Gln Xaa Gly Ile Asp Leu Glu Trp Phe Xaa Tyr Phe
130 135 140
Lys Xaa Glu Leu Thr Leu Xaa Xaa Xaa Glu Ser Ala Tyr Leu Xaa Xaa
145 150 155 160
Asn Asn Xaa Leu Val Xaa Xaa Gln Ile Lys Thr Gln Asn Lys Leu Ala
165 170 175
Leu Asp Leu Lys Gly Xaa Xaa Phe Val Leu Lys Thr Tyr Ile Tyr Pro
180 185 190
Ala Leu Lys Ser Xaa Ala Thr Gly Lys Ser Ile His Glu Leu Xaa Phe
195 200 205
Gly Ser Val Xaa Arg Leu Ser Xaa Xaa Xaa Pro Ser Ile Arg Xaa Pro
210 215 220
Leu Xaa Xaa Leu Glu Xaa Tyr Val Xaa Ser Arg Asn Xaa Xaa Xaa Xaa
225 230 235 240
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Leu Ser Pro Arg Leu Leu Ser Cys
245 250 255
Asp Leu Xaa Asp Pro Xaa Lys Ser Arg Val Lys Ile Tyr Leu Leu Glu
260 265 270
Arg Met Val Ser Leu Xaa Ala Met Glu Asp Leu Trp Thr Leu Gly Gly
275 280 285
Arg Arg Xaa Asp Xaa Ser Thr Xaa Xaa Gly Leu Xaa Met Ile Arg Glu
290 295 300
Leu Trp Xaa Leu Leu Xaa Xaa Pro Xaa Gly Leu Xaa Xaa Tyr Pro Xaa
305 310 315 320
Pro Tyr Leu Pro Leu Gly Xaa Ile Pro Xaa Glu Gln Leu Pro Ser Met
325 330 335
Ala Asn Tyr Thr Leu His His Xaa Asp Pro Xaa Pro Glu Pro Gln Val
340 345 350
Tyr Phe Thr Val Phe Gly Met Asn Asp Met Xaa Val Xaa Asp Ala Leu
355 360 365
Thr Thr Phe Phe Glu Arg Arg Gly Trp Xaa Xaa Met Ala Xaa Lys Tyr
370 375 380
Lys Ala Xaa Leu Xaa Xaa Ser Tyr Pro His Xaa Asp His Glu Xaa Leu
385 390 395 400
Asn Tyr Leu His Ala Tyr Ile Ser Phe Ser Tyr Arg Xaa Asn Lys Pro
405 410 415
Tyr Leu Ser Val Tyr Leu His Ser Phe Glu Thr Gly Asp Trp Pro Xaa
420 425 430
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Phe Asp Xaa Xaa Arg Arg Xaa Xaa Xaa
435 440 445
Xaa Xaa Lys
450
<210> 183
<211> 299
<212> PRT
<213> Artificial Sequence
<220>
<223> EasG consensus sequence 2
<220>
<221> misc_feature
<222> (15)..(15)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (21)..(22)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (24)..(26)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (37)..(38)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (40)..(41)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (46)..(48)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (55)..(55)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (60)..(60)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (63)..(70)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (73)..(73)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (77)..(77)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (82)..(82)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (86)..(86)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (100)..(100)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (124)..(124)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (133)..(134)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (137)..(137)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (139)..(139)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (152)..(154)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (157)..(157)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (160)..(160)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (162)..(163)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (181)..(182)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (196)..(196)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (198)..(198)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (201)..(201)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (236)..(236)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (240)..(240)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (243)..(244)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (246)..(246)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (250)..(250)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (252)..(252)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (254)..(254)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (262)..(262)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (265)..(265)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (273)..(275)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (278)..(278)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (282)..(283)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (292)..(294)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (296)..(297)
<223> Xaa can be any naturally occurring amino acid
<400> 183
Met Thr Ile Leu Leu Thr Gly Gly Arg Gly Lys Thr Ala Ser Xaa Ile
1 5 10 15
Ala Ser Leu Leu Xaa Xaa Ala Xaa Xaa Xaa Val Pro Phe Ile Val Ala
20 25 30
Ser Arg Ser Ser Xaa Xaa Ser Xaa Xaa Ser Pro Tyr Arg Xaa Xaa Xaa
35 40 45
Phe Asp Trp Leu Asp Glu Xaa Thr Phe Gly Asn Xaa Leu Ser Xaa Xaa
50 55 60
Xaa Xaa Xaa Xaa Xaa Xaa Gly Met Xaa Pro Ile Ser Xaa Val Trp Leu
65 70 75 80
Val Xaa Pro Pro Ile Xaa Asp Leu Ala Pro Pro Met Ile Lys Phe Ile
85 90 95
Asp Phe Ala Xaa Ser Arg Gly Val Lys Arg Phe Val Leu Leu Ser Ala
100 105 110
Ser Thr Ile Glu Lys Gly Gly Pro Ala Met Gly Xaa Val His Ala His
115 120 125
Leu Asp Ser Leu Xaa Xaa Gly Val Xaa Tyr Xaa Val Leu Arg Pro Thr
130 135 140
Trp Phe Met Glu Asn Phe Ser Xaa Xaa Xaa Glu Leu Xaa Trp Ile Xaa
145 150 155 160
Ile Xaa Xaa Glu Asn Lys Ile Tyr Ser Ala Thr Gly Asp Gly Lys Ile
165 170 175
Pro Phe Ile Ser Xaa Xaa Asp Ile Ala Arg Val Ala Phe Arg Ala Leu
180 185 190
Thr Asp Glu Xaa Ser Xaa Asn Thr Xaa Asp Tyr Val Leu Leu Gly Pro
195 200 205
Glu Leu Leu Thr Tyr Asp Asp Val Ala Glu Ile Leu Thr Thr Val Leu
210 215 220
Gly Arg Lys Ile Thr His Val Lys Leu Thr Glu Xaa Glu Leu Ala Xaa
225 230 235 240
Lys Leu Xaa Xaa Glu Xaa Gly Leu Pro Xaa Asp Xaa Ala Xaa Met Leu
245 250 255
Ala Ser Met Asp Thr Xaa Val Lys Xaa Gly Ala Glu Glu Arg Leu Asn
260 265 270
Xaa Xaa Xaa Val Lys Xaa Val Thr Gly Xaa Xaa Pro Arg Thr Phe Leu
275 280 285
Asp Phe Ala Xaa Xaa Xaa Lys Xaa Xaa Trp Leu
290 295
<210> 184
<211> 389
<212> PRT
<213> Artificial Sequence
<220>
<223> EasA consensus sequence
<220>
<221> misc_feature
<222> (7)..(7)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (10)..(10)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (13)..(13)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (15)..(16)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (34)..(34)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (38)..(38)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (44)..(44)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (63)..(63)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (79)..(79)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (88)..(88)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (117)..(119)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (121)..(121)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (135)..(135)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (148)..(149)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (163)..(163)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (166)..(166)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (186)..(186)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (190)..(190)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (194)..(194)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (237)..(237)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (250)..(250)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (256)..(261)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (279)..(282)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (286)..(288)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (290)..(290)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (297)..(307)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (309)..(310)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (319)..(319)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (325)..(325)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (329)..(330)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (333)..(333)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (335)..(336)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (341)..(341)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (358)..(358)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (361)..(361)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (374)..(374)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (376)..(376)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (386)..(386)
<223> Xaa can be any naturally occurring amino acid
<400> 184
Ser Ser Ser Lys Leu Phe Xaa Pro Leu Xaa Val Gly Xaa Arg Xaa Xaa
1 5 10 15
Leu Gln His Arg Ile Val Met Ala Pro Met Thr Arg Phe Arg Ala Asp
20 25 30
Asp Xaa Gly Val Pro Xaa Leu Pro Tyr Val Lys Xaa Tyr Tyr Ala Gln
35 40 45
Arg Ala Ser Val Pro Gly Thr Leu Leu Ile Thr Glu Ala Thr Xaa Ile
50 55 60
Ser Pro Arg Ala Gly Gly Phe Pro Asn Val Pro Gly Ile Trp Xaa Glu
65 70 75 80
Ala Gln Ile Ala Ala Trp Lys Xaa Val Val Asp Ala Val His Ala Lys
85 90 95
Gly Ser Phe Ile Phe Leu Gln Leu Trp Ala Thr Gly Arg Ala Ala Asp
100 105 110
Pro Asp Val Leu Xaa Xaa Xaa Gly Xaa Phe Asp Leu Val Ser Ser Ser
115 120 125
Ala Val Pro Val Ala Pro Xaa Gly Pro Val Pro Arg Ala Leu Thr Glu
130 135 140
Asp Glu Ile Xaa Xaa Tyr Ile Ala Asp Tyr Ala Gln Ala Ala Lys Asn
145 150 155 160
Ala Val Xaa Ala Gly Xaa Phe Asp Gly Val Glu Ile His Gly Ala Asn
165 170 175
Gly Tyr Leu Ile Asp Gln Phe Thr Gln Xaa Ser Cys Asn Xaa Arg Thr
180 185 190
Asp Xaa Trp Gly Gly Ser Ile Glu Asn Arg Ala Arg Phe Ala Leu Glu
195 200 205
Val Thr Arg Ala Val Val Asp Ala Val Gly Ala Asp Arg Val Gly Val
210 215 220
Lys Leu Ser Pro Trp Ser Thr Phe Gln Gly Met Gly Xaa Met Asp Asp
225 230 235 240
Leu Val Pro Gln Phe Glu Tyr Leu Ile Xaa Arg Leu Arg Glu Met Xaa
245 250 255
Xaa Xaa Xaa Xaa Xaa Leu Ala Tyr Leu His Leu Ala Asn Ser Arg Trp
260 265 270
Leu Glu Glu Glu Asp Pro Xaa Xaa Xaa Xaa His Pro Asp Xaa Xaa Xaa
275 280 285
Glu Xaa Phe Val Arg Met Trp Gly Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
290 295 300
Xaa Xaa Xaa Lys Xaa Xaa Pro Ile Leu Leu Ala Gly Gly Tyr Xaa Ala
305 310 315 320
Asp Ser Ala Lys Xaa Leu Val Asp Xaa Xaa Tyr Ser Xaa Asp Xaa Xaa
325 330 335
Asn Val Leu Val Xaa Phe Gly Arg His Phe Ile Ser Asn Pro Asp Leu
340 345 350
Pro Phe Arg Leu Lys Xaa Gly Ile Xaa Leu Gln Lys Tyr Asp Arg Asp
355 360 365
Thr Phe Tyr Ile Pro Xaa Ser Xaa Glu Gly Tyr Leu Asp Tyr Pro Phe
370 375 380
Ser Xaa Glu Phe Leu
385
<210> 185
<211> 606
<212> PRT
<213> Artificial Sequence
<220>
<223> EasE consensus sequence 2
<220>
<221> misc_feature
<222> (4)..(4)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (13)..(13)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (17)..(18)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (20)..(22)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (38)..(38)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (40)..(40)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (49)..(49)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (64)..(64)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (67)..(67)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (75)..(75)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (89)..(89)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (99)..(99)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (110)..(110)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (112)..(113)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (115)..(117)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (135)..(135)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (139)..(139)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (160)..(160)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (181)..(181)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (184)..(184)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (189)..(189)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (192)..(195)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (200)..(200)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (217)..(217)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (221)..(221)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (252)..(253)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (269)..(269)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (319)..(319)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (321)..(322)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (324)..(324)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (330)..(330)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (334)..(334)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (354)..(357)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (366)..(366)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (375)..(375)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (377)..(377)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (380)..(380)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (385)..(385)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (388)..(388)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (391)..(392)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (395)..(395)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (398)..(398)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (402)..(402)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (405)..(406)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (409)..(409)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (412)..(413)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (421)..(421)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (426)..(426)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (434)..(434)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (437)..(437)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (443)..(443)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (447)..(448)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (451)..(451)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (457)..(457)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (474)..(475)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (500)..(500)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (509)..(509)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (513)..(513)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (517)..(517)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (528)..(528)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (551)..(551)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (553)..(553)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (559)..(559)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (580)..(580)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (590)..(593)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (595)..(596)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (598)..(598)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (600)..(600)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (602)..(603)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (605)..(605)
<223> Xaa can be any naturally occurring amino acid
<400> 185
Leu Leu Thr Xaa Gly Leu Val Ala Leu Leu Ser Trp Xaa Ile Tyr Thr
1 5 10 15
Xaa Xaa Thr Xaa Xaa Xaa Ser Cys Arg Cys Arg Pro Trp Glu Ser Cys
20 25 30
Trp Pro Ser Glu Glu Xaa Trp Xaa Ser Leu Asn Thr Ser Ile Asp Gly
35 40 45
Xaa Leu Val Arg Leu Arg Pro Ile Ala His Val Cys His Gly Pro Xaa
50 55 60
Phe Asp Xaa Ser Ala Cys Asp Asn Leu Leu Xaa Leu Ser Arg Asp Ser
65 70 75 80
Gly Trp Arg Ala Ser Asn Pro Gly Xaa Leu Gln Asp Trp Val Trp Glu
85 90 95
Ala Gly Xaa Thr Ala Asn Glu Ser Cys Pro Val Gly Ser Xaa Arg Xaa
100 105 110
Xaa Ser Xaa Xaa Xaa Ser Cys His Gln Gly Arg Ile Pro Leu Tyr Ser
115 120 125
Ala Ala Val Glu Ser Thr Xaa Gln Val Gln Xaa Ala Val Arg Phe Ala
130 135 140
Arg Arg His Asn Leu Arg Leu Val Ile Arg Asn Thr Gly His Asp Xaa
145 150 155 160
Ala Gly Arg Ser Ser Ala Pro Asp Ser Phe Gln Ile His Thr His Arg
165 170 175
Leu Gln Glu Ile Xaa Phe His Xaa Asn Phe Arg Leu Xaa Gly Ser Xaa
180 185 190
Xaa Xaa Xaa Thr Ser Leu Gly Xaa Pro Ala Val Thr Val Gly Ala Gly
195 200 205
Val Met Met Gly Glu Leu Tyr Ala Xaa Gly Ala Arg Xaa Gly Tyr Met
210 215 220
Val Leu Gly Gly Glu Cys Pro Thr Val Gly Val Val Gly Gly Phe Leu
225 230 235 240
Gln Gly Gly Gly Val Ser Ser Phe Leu Ser Phe Xaa Xaa Gly Leu Ala
245 250 255
Val Asp Asn Val Leu Glu Tyr Glu Val Val Thr Ala Xaa Gly Glu Leu
260 265 270
Val Val Ala Asn Ala His Gln Asn Gln Asp Leu Phe Trp Ala Leu Arg
275 280 285
Gly Gly Gly Gly Gly Thr Phe Gly Val Val Thr Arg Ala Thr Met Arg
290 295 300
Val Phe Pro Asp Val Pro Val Val Val Ser Glu Ile Leu Leu Xaa Ala
305 310 315 320
Xaa Xaa Ala Xaa Ser Ser Phe Trp Thr Xaa Gly Leu Ser Xaa Leu Leu
325 330 335
Thr Ala Leu Gln Ser Leu Asn Gln Asp Asn Val Gly Gly Gln Leu Val
340 345 350
Ile Xaa Xaa Xaa Xaa Lys Asp Ala Val Gln Ala Ser Ile Xaa Leu Phe
355 360 365
Phe Leu Asn Met Thr Glu Xaa Ala Xaa Ile Asp Xaa Arg Met Lys Pro
370 375 380
Xaa Leu Thr Xaa Leu Ser Xaa Xaa Asn Ile Xaa Tyr Thr Xaa Ser Ser
385 390 395 400
Lys Xaa Leu Pro Xaa Xaa Ser Ser Xaa Leu Arg Xaa Xaa Pro Asp Ile
405 410 415
His Pro Asp Asn Xaa Tyr Gly Ile Leu Xaa Ser Ser Val Leu Ile Ser
420 425 430
Gln Xaa Leu Phe Xaa Ser Ser Glu Gly Pro Xaa Lys Val Ala Xaa Xaa
435 440 445
Leu Ala Xaa Leu Pro Met Ser Pro Xaa Asp Leu Leu Phe Thr Ser Asn
450 455 460
Leu Gly Gly Arg Val Ile Ala Asn Gly Xaa Xaa Glu Leu Ala Glu Thr
465 470 475 480
Ser Met His Pro Ala Trp Arg Ser Ala Ala Gln Leu Ile Asn Tyr Val
485 490 495
Arg Thr Val Xaa Glu Pro Ser Ile Glu Gly Lys Ala Xaa Ala Leu Glu
500 505 510
Xaa Leu Thr Asn Xaa Gln Met Pro Ile Leu Tyr Ala Ile Asp Pro Xaa
515 520 525
Phe Lys Leu Ser Tyr Arg Asn Leu Gly Asp Pro Asn Glu Lys Asp Phe
530 535 540
Gln Gln Val Tyr Trp Gly Xaa Asn Xaa Tyr Gly Arg Leu Ser Xaa Ile
545 550 555 560
Lys Lys Lys Trp Asp Pro Asp Asp Leu Phe Phe Ser Lys Leu Gly Val
565 570 575
Gly Ser Glu Xaa Trp Asp Ser Glu Gly Met Cys Arg Lys Xaa Xaa Xaa
580 585 590
Xaa Gln Xaa Xaa Leu Xaa Gln Xaa Leu Xaa Xaa Leu Xaa Ser
595 600 605
<210> 186
<211> 1692
<212> DNA
<213> Saccharomyces cerevisiae
<400> 186
atgagattaa gaaccgccat tgccacactg tgcctcacgg cttttacatc tgcaacttca 60
aacaatagct acatcgccac cgaccaaaca caaaatgcct ttaatgacac tcacttttgt 120
aaggtcgaca ggaatgatca cgttagtccc agttgtaacg taacattcaa tgaattaaat 180
gccataaatg aaaacattag agatgatctt tcggcgttat taaaatctga tttcttcaaa 240
tactttcggc tggatttata caagcaatgt tcattttggg acgccaacga tggtctgtgc 300
ttaaaccgcg cttgctctgt tgatgtcgta gaggactggg atacactgcc tgagtactgg 360
cagcctgaga tcttgggtag tttcaataat gatacaatga aggaagcgga tgatagcgat 420
gacgaatgta agttcttaga tcaactatgt caaaccagta aaaaacctgt agatatcgaa 480
gacaccatca actactgtga tgtaaatgac tttaacggta aaaacgccgt tctgattgat 540
ttaacagcaa atccggaacg atttacaggt tatggtggta agcaagctgg tcaaatttgg 600
tctactatct accaagacaa ctgttttaca attggcgaaa ctggtgaatc attggccaaa 660
gatgcatttt atagacttgt atccggtttc catgcctcta tcggtactca cttatcaaag 720
gaatatttga acacgaaaac tggtaaatgg gagcccaatc tggatttgtt tatggcaaga 780
atcgggaact ttcctgatag agtgacaaac atgtatttca attatgctgt tgtagctaag 840
gctctctgga aaattcaacc atatttacca gaattttcat tctgtgatct agtcaataaa 900
gaaatcaaaa acaaaatgga taacgttatt tcccagctgg acacaaaaat ttttaacgaa 960
gacttagttt ttgccaacga cctaagtttg actttgaagg acgaattcag atctcgcttc 1020
aagaatgtca cgaagattat ggattgtgtg caatgtgata gatgtagatt gtggggcaaa 1080
attcaaacta ccggttacgc aactgccttg aaaattttgt ttgaaatcaa cgacgctgat 1140
gaattcacca aacaacatat tgttggtaag ttaaccaaat atgagttgat tgcactatta 1200
cagactttcg gtagattatc tgaatctatt gaatctgtta acatgttcga aaaaatgtac 1260
gggaaaaggt taaacggttc tgaaaacagg ttaagctcat tcttccaaaa taacttcttc 1320
aacattttga aggaggcagg caaatcgatt cgttacacca tagagaacat caattccact 1380
aaagaaggaa agaaaaagac taacaattct caatcacatg tatttgatga tttaaaaatg 1440
cccaaagcag aaatagttcc aaggccctct aacggtacag taaataaatg gaagaaagct 1500
tggaatactg aagttaacaa cgttttagaa gcattcagat ttatttatag aagctatttg 1560
gatttaccca ggaacatctg ggaattatct ttgatgaagg tatacaaatt ttggaataaa 1620
ttcatcggtg ttgctgatta cgttagtgag gagacacgag agcctatttc ctataagcta 1680
gatatacaat aa 1692
<210> 187
<211> 563
<212> PRT
<213> Saccharomyces cerevisiae
<400> 187
Met Arg Leu Arg Thr Ala Ile Ala Thr Leu Cys Leu Thr Ala Phe Thr
1 5 10 15
Ser Ala Thr Ser Asn Asn Ser Tyr Ile Ala Thr Asp Gln Thr Gln Asn
20 25 30
Ala Phe Asn Asp Thr His Phe Cys Lys Val Asp Arg Asn Asp His Val
35 40 45
Ser Pro Ser Cys Asn Val Thr Phe Asn Glu Leu Asn Ala Ile Asn Glu
50 55 60
Asn Ile Arg Asp Asp Leu Ser Ala Leu Leu Lys Ser Asp Phe Phe Lys
65 70 75 80
Tyr Phe Arg Leu Asp Leu Tyr Lys Gln Cys Ser Phe Trp Asp Ala Asn
85 90 95
Asp Gly Leu Cys Leu Asn Arg Ala Cys Ser Val Asp Val Val Glu Asp
100 105 110
Trp Asp Thr Leu Pro Glu Tyr Trp Gln Pro Glu Ile Leu Gly Ser Phe
115 120 125
Asn Asn Asp Thr Met Lys Glu Ala Asp Asp Ser Asp Asp Glu Cys Lys
130 135 140
Phe Leu Asp Gln Leu Cys Gln Thr Ser Lys Lys Pro Val Asp Ile Glu
145 150 155 160
Asp Thr Ile Asn Tyr Cys Asp Val Asn Asp Phe Asn Gly Lys Asn Ala
165 170 175
Val Leu Ile Asp Leu Thr Ala Asn Pro Glu Arg Phe Thr Gly Tyr Gly
180 185 190
Gly Lys Gln Ala Gly Gln Ile Trp Ser Thr Ile Tyr Gln Asp Asn Cys
195 200 205
Phe Thr Ile Gly Glu Thr Gly Glu Ser Leu Ala Lys Asp Ala Phe Tyr
210 215 220
Arg Leu Val Ser Gly Phe His Ala Ser Ile Gly Thr His Leu Ser Lys
225 230 235 240
Glu Tyr Leu Asn Thr Lys Thr Gly Lys Trp Glu Pro Asn Leu Asp Leu
245 250 255
Phe Met Ala Arg Ile Gly Asn Phe Pro Asp Arg Val Thr Asn Met Tyr
260 265 270
Phe Asn Tyr Ala Val Val Ala Lys Ala Leu Trp Lys Ile Gln Pro Tyr
275 280 285
Leu Pro Glu Phe Ser Phe Cys Asp Leu Val Asn Lys Glu Ile Lys Asn
290 295 300
Lys Met Asp Asn Val Ile Ser Gln Leu Asp Thr Lys Ile Phe Asn Glu
305 310 315 320
Asp Leu Val Phe Ala Asn Asp Leu Ser Leu Thr Leu Lys Asp Glu Phe
325 330 335
Arg Ser Arg Phe Lys Asn Val Thr Lys Ile Met Asp Cys Val Gln Cys
340 345 350
Asp Arg Cys Arg Leu Trp Gly Lys Ile Gln Thr Thr Gly Tyr Ala Thr
355 360 365
Ala Leu Lys Ile Leu Phe Glu Ile Asn Asp Ala Asp Glu Phe Thr Lys
370 375 380
Gln His Ile Val Gly Lys Leu Thr Lys Tyr Glu Leu Ile Ala Leu Leu
385 390 395 400
Gln Thr Phe Gly Arg Leu Ser Glu Ser Ile Glu Ser Val Asn Met Phe
405 410 415
Glu Lys Met Tyr Gly Lys Arg Leu Asn Gly Ser Glu Asn Arg Leu Ser
420 425 430
Ser Phe Phe Gln Asn Asn Phe Phe Asn Ile Leu Lys Glu Ala Gly Lys
435 440 445
Ser Ile Arg Tyr Thr Ile Glu Asn Ile Asn Ser Thr Lys Glu Gly Lys
450 455 460
Lys Lys Thr Asn Asn Ser Gln Ser His Val Phe Asp Asp Leu Lys Met
465 470 475 480
Pro Lys Ala Glu Ile Val Pro Arg Pro Ser Asn Gly Thr Val Asn Lys
485 490 495
Trp Lys Lys Ala Trp Asn Thr Glu Val Asn Asn Val Leu Glu Ala Phe
500 505 510
Arg Phe Ile Tyr Arg Ser Tyr Leu Asp Leu Pro Arg Asn Ile Trp Glu
515 520 525
Leu Ser Leu Met Lys Val Tyr Lys Phe Trp Asn Lys Phe Ile Gly Val
530 535 540
Ala Asp Tyr Val Ser Glu Glu Thr Arg Glu Pro Ile Ser Tyr Lys Leu
545 550 555 560
Asp Ile Gln
<210> 188
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> EasD cofactor-binding motif
<220>
<221> misc_feature
<222> (3)..(5)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (7)..(7)
<223> Xaa can be any naturally occurring amino acid
<400> 188
Thr Gly Xaa Xaa Xaa Gly Xaa Gly
1 5
<210> 189
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> EasD active site motif
<220>
<221> misc_feature
<222> (2)..(4)
<223> Xaa can be any naturally occurring amino acid
<400> 189
Tyr Xaa Xaa Xaa Lys
1 5
<210> 190
<211> 430
<212> PRT
<213> Artificial Sequence
<220>
<223> DmaW_consensus sequence No 3
<220>
<221> misc_feature
<222> (2)..(2)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (4)..(4)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (6)..(6)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (8)..(9)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (13)..(13)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (17)..(18)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (24)..(24)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (27)..(27)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (39)..(39)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (47)..(48)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (50)..(50)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (53)..(53)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (72)..(73)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (75)..(75)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (96)..(96)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (98)..(98)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (113)..(113)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (120)..(120)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (124)..(124)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (128)..(128)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (130)..(132)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (134)..(134)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (142)..(142)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (146)..(146)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (151)..(153)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (159)..(160)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (163)..(163)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (166)..(167)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (182)..(183)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (197)..(197)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (207)..(207)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (212)..(212)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (216)..(218)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (223)..(223)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (226)..(227)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (230)..(230)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (233)..(233)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (237)..(258)
<223> the stretch of X between amino acid position 237 and 258 can be
between 4 X and 12 X
<220>
<221> misc_feature
<222> (259)..(259)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (262)..(262)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (278)..(278)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (291)..(291)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (293)..(293)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (296)..(297)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (300)..(300)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (307)..(307)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (310)..(311)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (313)..(313)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (316)..(317)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (320)..(320)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (327)..(327)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (330)..(330)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (344)..(344)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (347)..(347)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (363)..(363)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (365)..(365)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (378)..(379)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (382)..(382)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (387)..(387)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (389)..(390)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (395)..(395)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (399)..(399)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (413)..(413)
<223> Xaa can be any naturally occurring amino acid
<400> 190
Met Xaa Thr Xaa Asn Xaa Ser Xaa Xaa Glu Val Tyr Xaa Thr Leu Ser
1 5 10 15
Xaa Xaa Phe Asp Phe Pro Asn Xaa Asp Gln Xaa Leu Trp Trp His Ser
20 25 30
Thr Ala Pro Met Phe Ala Xaa Met Leu Gln Thr Ala Asn Tyr Xaa Xaa
35 40 45
His Xaa Gln Tyr Xaa His Leu Gly Ile Tyr Lys Lys His Val Ile Pro
50 55 60
Phe Leu Gly Val Tyr Pro Thr Xaa Xaa Lys Xaa Arg Trp Leu Ser Ile
65 70 75 80
Leu Thr Arg Tyr Gly Thr Pro Phe Glu Leu Ser Leu Asn Cys Ser Xaa
85 90 95
Ser Xaa Val Arg Tyr Thr Tyr Glu Pro Ile Asn Ala Ala Thr Gly Thr
100 105 110
Xaa Lys Asp Pro Phe Asn Thr Xaa Ala Ile Trp Xaa Ser Leu Gln Xaa
115 120 125
Leu Xaa Xaa Xaa Gln Xaa Gly Ile Asp Leu Glu Trp Phe Xaa Tyr Phe
130 135 140
Lys Xaa Glu Leu Thr Leu Xaa Xaa Xaa Glu Ser Ala Tyr Leu Xaa Xaa
145 150 155 160
Asn Asn Xaa Leu Val Xaa Xaa Gln Ile Lys Thr Gln Asn Lys Leu Ala
165 170 175
Leu Asp Leu Lys Gly Xaa Xaa Phe Val Leu Lys Thr Tyr Ile Tyr Pro
180 185 190
Ala Leu Lys Ser Xaa Ala Thr Gly Lys Ser Ile His Glu Leu Xaa Phe
195 200 205
Gly Ser Val Xaa Arg Leu Ser Xaa Xaa Xaa Pro Ser Ile Arg Xaa Pro
210 215 220
Leu Xaa Xaa Leu Glu Xaa Tyr Val Xaa Ser Arg Asn Xaa Xaa Xaa Xaa
225 230 235 240
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Leu Ser Pro Arg Leu Leu Ser Cys
245 250 255
Asp Leu Xaa Asp Pro Xaa Lys Ser Arg Val Lys Ile Tyr Leu Leu Glu
260 265 270
Arg Met Val Ser Leu Xaa Ala Met Glu Asp Leu Trp Thr Leu Gly Gly
275 280 285
Arg Arg Xaa Asp Xaa Ser Thr Xaa Xaa Gly Leu Xaa Met Ile Arg Glu
290 295 300
Leu Trp Xaa Leu Leu Xaa Xaa Pro Xaa Gly Leu Xaa Xaa Tyr Pro Xaa
305 310 315 320
Pro Tyr Leu Pro Leu Gly Xaa Ile Pro Xaa Glu Gln Leu Pro Ser Met
325 330 335
Ala Asn Tyr Thr Leu His His Xaa Asp Pro Xaa Pro Glu Pro Gln Val
340 345 350
Tyr Phe Thr Val Phe Gly Met Asn Asp Met Xaa Val Xaa Asp Ala Leu
355 360 365
Thr Thr Phe Phe Glu Arg Arg Gly Trp Xaa Xaa Met Ala Xaa Lys Tyr
370 375 380
Lys Ala Xaa Leu Xaa Xaa Ser Tyr Pro His Xaa Asp His Glu Xaa Leu
385 390 395 400
Asn Tyr Leu His Ala Tyr Ile Ser Phe Ser Tyr Arg Xaa Asn Lys Pro
405 410 415
Tyr Leu Ser Val Tyr Leu His Ser Phe Glu Thr Gly Asp Trp
420 425 430
<210> 191
<211> 566
<212> PRT
<213> Artificial Sequence
<220>
<223> EasE_ core consensus sequence
<220>
<221> misc_feature
<222> (15)..(15)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (17)..(17)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (26)..(26)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (41)..(41)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (44)..(44)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (52)..(52)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (66)..(66)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (76)..(76)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (87)..(87)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (88)..(94)
<223> the stretch of X between amino acid position 88 and 94 can be
between 2 X and 6 X
<220>
<221> misc_feature
<222> (112)..(112)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (116)..(116)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (137)..(137)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (158)..(158)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (161)..(161)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (166)..(166)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (169)..(172)
<223> the stretch of X between amino acid position 169 and 172 can be
between 1 X and 4 X
<220>
<221> misc_feature
<222> (177)..(177)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (194)..(194)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (198)..(198)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (229)..(230)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (246)..(246)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (296)..(296)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (298)..(299)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (301)..(301)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (307)..(307)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (311)..(311)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (331)..(334)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (343)..(343)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (352)..(352)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (354)..(354)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (357)..(357)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (362)..(362)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (365)..(365)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (368)..(369)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (372)..(372)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (375)..(375)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (379)..(379)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (382)..(383)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (386)..(386)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (389)..(390)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (398)..(398)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (403)..(403)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (411)..(411)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (414)..(414)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (420)..(420)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (424)..(425)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (428)..(428)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (434)..(434)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (451)..(452)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (477)..(477)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (486)..(486)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (490)..(490)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (494)..(494)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (505)..(505)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (528)..(528)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (530)..(530)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (536)..(536)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (557)..(557)
<223> Xaa can be any naturally occurring amino acid
<400> 191
Cys Arg Cys Arg Pro Trp Glu Ser Cys Trp Pro Ser Glu Glu Xaa Trp
1 5 10 15
Xaa Ser Leu Asn Thr Ser Ile Asp Gly Xaa Leu Val Arg Leu Arg Pro
20 25 30
Ile Ala His Val Cys His Gly Pro Xaa Phe Asp Xaa Ser Ala Cys Asp
35 40 45
Asn Leu Leu Xaa Leu Ser Arg Asp Ser Gly Trp Arg Ala Ser Asn Pro
50 55 60
Gly Xaa Leu Gln Asp Trp Val Trp Glu Ala Gly Xaa Thr Ala Asn Glu
65 70 75 80
Ser Cys Pro Val Gly Ser Xaa Arg Xaa Xaa Xaa Xaa Xaa Xaa Ser Cys
85 90 95
His Gln Gly Arg Ile Pro Leu Tyr Ser Ala Ala Val Glu Ser Thr Xaa
100 105 110
Gln Val Gln Xaa Ala Val Arg Phe Ala Arg Arg His Asn Leu Arg Leu
115 120 125
Val Ile Arg Asn Thr Gly His Asp Xaa Ala Gly Arg Ser Ser Ala Pro
130 135 140
Asp Ser Phe Gln Ile His Thr His Arg Leu Gln Glu Ile Xaa Phe His
145 150 155 160
Xaa Asn Phe Arg Leu Xaa Gly Ser Xaa Xaa Xaa Xaa Thr Ser Leu Gly
165 170 175
Xaa Pro Ala Val Thr Val Gly Ala Gly Val Met Met Gly Glu Leu Tyr
180 185 190
Ala Xaa Gly Ala Arg Xaa Gly Tyr Met Val Leu Gly Gly Glu Cys Pro
195 200 205
Thr Val Gly Val Val Gly Gly Phe Leu Gln Gly Gly Gly Val Ser Ser
210 215 220
Phe Leu Ser Phe Xaa Xaa Gly Leu Ala Val Asp Asn Val Leu Glu Tyr
225 230 235 240
Glu Val Val Thr Ala Xaa Gly Glu Leu Val Val Ala Asn Ala His Gln
245 250 255
Asn Gln Asp Leu Phe Trp Ala Leu Arg Gly Gly Gly Gly Gly Thr Phe
260 265 270
Gly Val Val Thr Arg Ala Thr Met Arg Val Phe Pro Asp Val Pro Val
275 280 285
Val Val Ser Glu Ile Leu Leu Xaa Ala Xaa Xaa Ala Xaa Ser Ser Phe
290 295 300
Trp Thr Xaa Gly Leu Ser Xaa Leu Leu Thr Ala Leu Gln Ser Leu Asn
305 310 315 320
Gln Asp Asn Val Gly Gly Gln Leu Val Ile Xaa Xaa Xaa Xaa Lys Asp
325 330 335
Ala Val Gln Ala Ser Ile Xaa Leu Phe Phe Leu Asn Met Thr Glu Xaa
340 345 350
Ala Xaa Ile Asp Xaa Arg Met Lys Pro Xaa Leu Thr Xaa Leu Ser Xaa
355 360 365
Xaa Asn Ile Xaa Tyr Thr Xaa Ser Ser Lys Xaa Leu Pro Xaa Xaa Ser
370 375 380
Ser Xaa Leu Arg Xaa Xaa Pro Asp Ile His Pro Asp Asn Xaa Tyr Gly
385 390 395 400
Ile Leu Xaa Ser Ser Val Leu Ile Ser Gln Xaa Leu Phe Xaa Ser Ser
405 410 415
Glu Gly Pro Xaa Lys Val Ala Xaa Xaa Leu Ala Xaa Leu Pro Met Ser
420 425 430
Pro Xaa Asp Leu Leu Phe Thr Ser Asn Leu Gly Gly Arg Val Ile Ala
435 440 445
Asn Gly Xaa Xaa Glu Leu Ala Glu Thr Ser Met His Pro Ala Trp Arg
450 455 460
Ser Ala Ala Gln Leu Ile Asn Tyr Val Arg Thr Val Xaa Glu Pro Ser
465 470 475 480
Ile Glu Gly Lys Ala Xaa Ala Leu Glu Xaa Leu Thr Asn Xaa Gln Met
485 490 495
Pro Ile Leu Tyr Ala Ile Asp Pro Xaa Phe Lys Leu Ser Tyr Arg Asn
500 505 510
Leu Gly Asp Pro Asn Glu Lys Asp Phe Gln Gln Val Tyr Trp Gly Xaa
515 520 525
Asn Xaa Tyr Gly Arg Leu Ser Xaa Ile Lys Lys Lys Trp Asp Pro Asp
530 535 540
Asp Leu Phe Phe Ser Lys Leu Gly Val Gly Ser Glu Xaa Trp Asp Ser
545 550 555 560
Glu Gly Met Cys Arg Lys
565
<210> 192
<211> 314
<212> PRT
<213> Artificial Sequence
<220>
<223> EasC core consensus sequence
<220>
<221> misc_feature
<222> (29)..(29)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (36)..(36)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (48)..(48)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (79)..(79)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (87)..(87)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (90)..(90)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (103)..(103)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (117)..(117)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (128)..(128)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (138)..(138)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (148)..(148)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (185)..(196)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (200)..(200)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (230)..(230)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (247)..(247)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (250)..(250)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (253)..(254)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (259)..(259)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (270)..(270)
<223> Xaa can be any naturally occurring amino acid
<400> 192
Leu Ala His Phe Asn Arg Glu Lys Ile Pro Glu Arg Ala Val His Ala
1 5 10 15
Lys Gly Ala Gly Ala Tyr Gly Glu Phe Glu Val Thr Xaa Asp Ile Ser
20 25 30
Asp Ile Cys Xaa Ile Asp Met Leu Leu Gly Val Gly Lys Lys Thr Xaa
35 40 45
Cys Val Thr Arg Phe Ser Thr Thr Gly Leu Glu Arg Gly Ser Ala Asp
50 55 60
Ser Val Arg Asp Leu Lys Gly Met Ala Val Lys Phe Tyr Thr Xaa Glu
65 70 75 80
Gly Asn Trp Asp Trp Val Xaa Leu Asn Xaa Pro Met Phe Phe Ile Arg
85 90 95
Asp Pro Ser Lys Phe Pro Xaa Leu Ile His Ala Gln Arg Arg Asp Pro
100 105 110
Gln Thr Asn Leu Xaa Asn Pro Ser Met Phe Trp Asp Phe Val Thr Xaa
115 120 125
Asn His Glu Ala Leu His Met Val Met Xaa Gln Phe Ser Asp Phe Gly
130 135 140
Thr Met Phe Xaa Tyr Arg Ser Met Ser Gly Tyr Val Gly His Ala Tyr
145 150 155 160
Lys Trp Val Met Pro Asp Gly Ser Phe Lys Tyr Val His Ile Phe Leu
165 170 175
Ser Ser Asp Gln Gly Pro Asn Phe Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
180 185 190
Xaa Xaa Xaa Xaa Asp Pro Asp Xaa Ala Thr Arg Asp Leu Tyr Glu Ala
195 200 205
Ile Glu Arg Gly Glu Tyr Pro Ser Trp Thr Ala Asn Val Gln Val Val
210 215 220
Asp Pro Glu Asp Ala Xaa Lys Leu Gly Phe Asn Ile Leu Asp Val Thr
225 230 235 240
Lys His Trp Asn Leu Gly Xaa Tyr Pro Xaa Asp Leu Xaa Xaa Ile Pro
245 250 255
Ser Arg Xaa Phe Gly Lys Leu Thr Leu Asn Lys Asn Pro Xaa Asn Tyr
260 265 270
Phe Ala Glu Ile Glu Gln Leu Ala Phe Ser Pro Ser His Leu Val Pro
275 280 285
Gly Val Glu Pro Ser Glu Asp Pro Ile Leu Gln Ala Arg Leu Phe Ala
290 295 300
Tyr Pro Asp Ala Gln Arg Tyr Arg Leu Gly
305 310
Claims (59)
- a) 적어도 하나의 EasH 활성, 또는
b) 적어도 하나의 EasH 및 EasD 활성, 또는
c) 적어도 하나의 EasH 및 EasA 리덕타제 활성, 또는
d) 적어도 하나의 EasH, EasD 및 EasA 활성, 또는
e) 적어도 하나의 EasH 및 EasD, EasA 리덕타제 및 EasG 활성, 또는
f) 적어도 하나의 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 및 EasG 활성, 또는
g) 적어도 하나의 EasD, EasA 및 EasG 활성, 또는
h) 적어도 하나의 DmaW, EasF, EasE, EasC, EasD, EasA 리덕타제 및 EasG 활성, 또는
i) 적어도 하나의 DmaW, EasF, EasE, EasC, EasD, EasA 이소머라제 및 EasG 활성, 또는
j) 적어도 하나의 DmaW, EasF, EasE 및 EasC 활성, 또는
k) a) 내지 j) 중 적어도 2개의 조합
을 포함하고, 천연 맥각 알칼로이드 생산자 유기체가 아닌 재조합 미생물. - DmaW, EasF, EasE, EasC, EasD, EasH, EasA 및 EasG 활성으로 이루어지는 활성의 군으로부터 선택된 적어도 하나의 상향조절된 활성을 갖고, 여기서 EasA 활성은 EasA 리덕타제, EasA 이소머라제 또는 EasA 리덕타제 및 EasA 이소머라제 활성인 재조합 천연 맥각 알칼로이드 생산자 유기체.
- 제2항에 있어서, 적어도 하나의 상향조절된 활성이
a) 적어도 하나의 EasH 활성, 또는
b) 적어도 하나의 EasH 및 EasD, 또는
c) 적어도 하나의 EasH 및 EasA 리덕타제 활성, 또는
d) 적어도 하나의 EasH, EasD 및 EasA 활성, 또는
e) 적어도 하나의 EasH 및 EasD, EasA 리덕타제 및 EasG 활성, 또는
f) 적어도 하나의 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 및 EasG 활성, 또는
g) 적어도 하나의 EasD, EasA 및 EasG 활성, 또는
h) 적어도 하나의 DmaW, EasF, EasE, EasC, EasD, EasA 리덕타제 및 EasG 활성, 또는
i) 적어도 하나의 DmaW, EasF, EasE, EasC, EasD, EasA 이소머라제 및 EasG 활성, 또는
j) 적어도 하나의 DmaW, EasF, EasE 및 EasC 활성
의 군 중의 하나 이상의 활성으로부터 선택되는 것인 재조합 천연 맥각 알칼로이드 생산자 유기체. - 제2항 또는 제3항에 있어서, EasD, EasH, EasA 리덕타제, EasA 이소머라제 및 EasG 활성의 군으로부터 선택된 적어도 하나의 하향조절된 활성을 갖는 재조합 천연 맥각 알칼로이드 생산자 유기체.
- 제1항 내지 제4항 중 어느 한 항에 있어서, DMAPP의 확대된 공급 및/또는 Me-DMAT의 확대된 공급을 포함하는, 바람직하게는 적어도 하나의 하향조절된 ERG9 또는 ERG20 활성, 또는 적어도 하나의 상향조절된 HMG-CoA 리덕타제 활성을 포함하는, 또는 적어도 하나의 하향조절된 ERG9 또는 ERG20 활성 및 적어도 하나의 상향조절된 HMG-CoA 리덕타제 활성을 포함하는 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체.
- 제1항 내지 제5항 중 어느 한 항에 있어서,
a) 적어도 하나의 EasH 활성, 또는
b) 적어도 하나의 EasH 및 EasD 활성, 또는
c) 적어도 하나의 EasA 리덕타제 또는 적어도 하나의 EasA 이소머라제 활성, 또는
d) 적어도 하나의 EasD, EasA 이소머라제 및 EasG 활성, 또는
e) 적어도 하나의 EasD, EasA 리덕타제 및 EasG 활성, 또는
f) 적어도 하나의 EasH, EasD, EasA 리덕타제, EasA 이소머라제 및 EasG 활성, 또는
g) 적어도 하나의 EasH, EasD, EasA 리덕타제 및 EasG 활성, 또는
h) 적어도 하나의 EasH, EasD, EasA 이소머라제 및 EasG 활성, 또는
i) 적어도 하나의 DmaW, EasF, EasE 및 EasC 활성, 또는
j) a) 내지 i) 중 적어도 2개의 조합
을 코딩하는 적어도 하나의 재조합 폴리뉴클레오티드를 포함하는 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체. - a) 적어도 하나의 EasH 활성, 또는
b) 적어도 하나의 EasH 및 EasD 활성, 또는
c) 적어도 하나의 EasA 리덕타제 또는 적어도 하나의 EasA 이소머라제 활성, 또는
d) 적어도 하나의 EasD, EasA 이소머라제 및 EasG 활성, 또는
e) 적어도 하나의 EasD, EasA 리덕타제 및 EasG 활성, 또는
f) 적어도 하나의 EasH, EasD, EasA 리덕타제, EasA 이소머라제 및 EasG 활성, 또는
g) 적어도 하나의 EasH, EasD, EasA 리덕타제 및 EasG 활성, 또는
h) 적어도 하나의 EasH, EasD, EasA 이소머라제 및 EasG 활성, 또는
i) 적어도 하나의 DmaW, EasF, EasE 및 EasC 활성, 또는
j) a) 내지 i) 중 적어도 2개의 조합
을 코딩하는 재조합 폴리뉴클레오티드. - 제1항 내지 제7항 중 어느 한 항에 있어서, DmaW 활성이
a) 서열 145 및 146에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
b) 서열 147 및/또는 190에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
c) 서열 1, 19, 20, 179, 및/또는 180에 적어도 40% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 1, 19, 20, 21, 22, 23, 24, 25, 26, 27, 및/또는 28에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
e) 서열 102, 122, 123에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
f) 서열 129 및/또는 137에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는,
g) 서열 102, 122, 123, 129 및/또는 137에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
h) 엄격한 조건 하에 서열 102, 122, 123, 129 및/또는 137에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
i) a) 내지 h) 중 적어도 2개, 바람직하게는 적어도 b) 및 c)에 따른 폴리펩티드, 또는
j) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 i) 중 적어도 하나에 따른 폴리펩티드
에 의한 것인 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드. - 제1항 내지 제8항 중 어느 한 항에 있어서, EasF 활성이
a) 서열 154에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
b) 서열 6, 75 및/또는 76에 적어도 40% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
c) 서열 6, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84 및/또는 85에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 107 및/또는 120에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
e) 서열 134 및/또는 142에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는,
f) 서열 107, 120, 134 및/또는 142에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
g) 엄격한 조건 하에 서열 107, 120, 134 및/또는 142에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
h) a) 내지 g) 중 적어도 2개, 바람직하게는 적어도 a) 및 b)에 따른 폴리펩티드 또는
i) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 h) 중 적어도 하나에 따른 폴리펩티드
에 의한 것인 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드. - 제1항 내지 제9항 중 어느 한 항에 있어서, EasE 활성이
a) 서열 153, 185 및/또는 191에 적어도 90% 서열 동일성, 바람직하게는 서열 191에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는,
b) 서열 5 및/또는 64에 적어도 40% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
c) 서열 5, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73 및/또는 74에 적어도 90% 서열 동일성, 또는 서열 5, 64, 175, 66, 67, 68, 69, 176, 71, 177, 73 및/또는 178에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 106에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
e) 서열 133 및/또는 141에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는,
f) 서열 106, 133 또는 141에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
g) 엄격한 조건 하에 서열 106, 133 또는 141에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
h) a) 내지 g) 중 적어도 2개, 바람직하게는 적어도 a) 및 b)에 따른 폴리펩티드 또는
i) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 h) 중 적어도 하나에 따른 폴리펩티드
에 의한 것인 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드. - 제1항 내지 제10항 중 어느 한 항에 있어서, EasC 활성이
a) 서열 151 및/또는 192에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는,
b) 서열 3 및/또는 41에 적어도 40% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
c) 서열 3, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51 및/또는 52에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 104에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
e) 서열 131 및/또는 139에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는,
f) 서열 104, 131 및/또는 139에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
g) 엄격한 조건 하에 서열 104, 131 및/또는 139에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
h) a) 내지 g) 중 적어도 2개, 바람직하게는 적어도 a) 및 b)에 따른 폴리펩티드, 또는
i) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 h) 중 적어도 하나에 따른 폴리펩티드
에 의한 것인 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드. - 제1항 내지 제11항 중 어느 한 항에 있어서, EasD 활성이
a) 서열 152에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는,
b) 서열 4 및/또는 53에 적어도 40% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
c) 서열 4, 53, 54, 55, 56, 57, 58, 59, 60, 62 및/또는 63에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 105에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
e) 서열 132 및/또는 140에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
f) 서열 105, 132 및/또는 140에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드 또는
g) 엄격한 조건 하에 서열 105, 132 및/또는 140에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드 또는
h) a) 내지 g) 중 적어도 2개, 바람직하게는 적어도 a) 및 b)에 따른 폴리펩티드 또는
i) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 h) 중 적어도 하나에 따른 폴리펩티드
에 의한 것인 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드. - 제1항 내지 제12항 중 어느 한 항에 있어서, EasH 활성이
a) 서열 156에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
b) 서열 8 및/또는 95에 적어도 60% 서열 동일성을 갖고 서열 156에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
c) 서열 8 및/또는 95에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 109 및/또는 157에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
e) 서열 109, 136 또는 144 및/또는 157에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는,
f) 서열 109, 136, 144 및/또는 157에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
g) 엄격한 조건 하에 서열 109, 136, 144 및/또는 157에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
h) a) 내지 g) 중 적어도 2개, 바람직하게는 적어도 a) 및 b)에 따른 폴리펩티드 또는
i) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 h) 중 적어도 하나에 따른 폴리펩티드
에 의한 것인 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드. - 제1항 내지 제13항 중 어느 한 항에 있어서, EasA 리덕타제 활성이
a) 서열 148, 149 및 150에 적어도 90% 서열 동일성을 갖고 서열 149의 아미노산 위치 18에 티로신을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
b) 서열 193, 194 및 195에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 194의 아미노산 위치 18에 티로신을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
c) 서열 2 및/또는 31에 적어도 40% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 2, 31, 32, 33, 34, 35, 36, 37, 38, 39 및/또는 40에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
e) 서열 103에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
f) 서열 130 및/또는 138에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는,
g) 서열 103, 130 및/또는 138에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
h) 엄격한 조건 하에 서열 103, 130 및/또는 138에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
i) a) 내지 h) 중 적어도 2개, 바람직하게는 적어도 b) 및 c)에 따른 폴리펩티드, 또는
j) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 i) 중 적어도 하나에 따른 폴리펩티드
에 의한 것인 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드. - 제1항 내지 제14항 중 어느 한 항에 있어서, EasA 이소머라제 활성이
a) 서열 148, 149 및 150에 적어도 90% 서열 동일성을 갖고 서열 149의 아미노산 위치 18에 페닐알라닌을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
b) 서열 193, 194 및 195에 적어도 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 194의 아미노산 위치 18에 페닐알라닌을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
c) 서열 2 및/또는 31에 적어도 40% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 2, 31, 32, 33, 34, 35, 36, 37, 38, 39 및/또는 40에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
e) 서열 103에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
f) 서열 130 및/또는 138에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는,
g) 서열 103, 130 및/또는 138에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
h) 엄격한 조건 하에 서열 103, 130 및/또는 138에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
i) a) 내지 g) 중 적어도 2개, 바람직하게는 적어도 a) 및 b)에 따른 폴리펩티드, 또는
j) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 h) 중 적어도 하나에 따른 폴리펩티드
에 의한 것인 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드. - 제1항 내지 제15항 중 어느 한 항에 있어서, EasG 활성이
a) 서열 155 및/또는 183에 적어도 90% 서열 동일성, 바람직하게는 서열 183에 적어도 90% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
b) 서열 7 및/또는 86에 적어도 39% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
c) 서열 7, 86, 87, 88, 89, 90, 181, 92, 93 및/또는 94에 적어도 70% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드, 또는
d) 서열 108에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
e) 서열 135 및/또는 143에 적어도 70% 서열 동일성을 갖는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
f) 서열 108, 135 및/또는 143에 의해 설명되는 서열의 군으로부터 선택되는 폴리뉴클레오티드 서열 중의 적어도 15개의 연속적인 뉴클레오티드를 각각 포함하는 2개의 PCR-프라이머를 사용하여 얻을 수 있는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
g) 엄격한 조건 하에 서열 108, 135 및/또는 143에 혼성화하는 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드, 또는
h) a) 내지 g) 중 적어도 2개, 바람직하게는 적어도 a) 및 b)에 따른 폴리펩티드, 또는
i) 천연 맥각 알칼로이드 생산자 유기체로부터 얻을 수 있는, a) 내지 h) 중 적어도 하나에 따른 폴리펩티드
에 의한 것인 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드. - 제1항 내지 제6항, 및 제8항 내지 제16항 중 어느 한 항에 있어서, 효모이고,
a. 서열 8 및/또는 95에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 8 및/또는 95에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 156에 적어도 90%, 바람직하게는 적어도 95% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 더 바람직하게는 서열 8 및/또는 95에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 8에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, EasH 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트,
b. 서열 2, 31, 32, 33, 34, 35, 36, 37, 38, 39 및/또는 40에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 2, 31, 32, 33, 34, 35, 36, 37, 38, 39 및/또는 40에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 148, 149 및 150에 적어도 95%, 바람직하게는 100% 서열 동일성을 갖고 서열 149의 아미노산 위치 18에 티로신을 갖는 아미노산 서열을 포함하는, EasA 리덕타제 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트,
c. 서열 7, 86, 87, 88, 89, 90, 181, 92, 93 및/또는 94에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 7, 86, 87, 88, 89, 90, 181, 92, 93 및/또는 94에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 183에 적어도 90%, 바람직하게는 적어도 95% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 바람직하게는 서열 7, 86, 87, 88, 89, 90, 181, 92, 93 및/또는 94에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 10a 내지 10c에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, EasG 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트,
d. 서열 4, 53, 54, 55, 56, 57, 58, 59, 60, 62 및/또는 63에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 4, 53, 54, 55, 56, 57, 58, 59, 60, 62 및/또는 63에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 152에 적어도 80%, 바람직하게는 적어도 81% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 바람직하게는 서열 4, 53, 54, 55, 56, 57, 58, 59, 60, 62 및/또는 63에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 7a 내지 7b에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, EasD 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트,
e. 서열 3 또는 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51 및/또는 52에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 3 또는 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51 및/또는 52에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 192에 적어도 85%, 바람직하게는 적어도 87% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 바람직하게는 서열 3 또는 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51 및/또는 52에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 6a 내지 6d에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, EasC 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트,
f. 서열 5, 64, 175, 66, 67, 68, 69, 176, 71, 177, 73 및/또는 178에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 5, 64, 175, 66, 67, 68, 69, 176, 71, 177, 73 및/또는 178에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 191에 적어도 70%, 바람직하게는 적어도 73% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 바람직하게는 서열 5, 64, 175, 66, 67, 68, 69, 176, 71, 177, 73 및/또는 178에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 5a 내지 5e에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, EasE 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트,
g. 서열 6, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84 및/또는 85에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 6, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84 및/또는 85에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 154에 적어도 75%, 바람직하게는 적어도 79% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 바람직하게는 서열 6, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84 및/또는 85에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 4a 내지 4c에 제시된 정렬에 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, EasF 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트,
h. 서열 1, 19, 20, 21, 22, 23, 24, 25, 26, 27 및/또는 28에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖는 아미노산 서열을 포함하는, 바람직하게는 서열 1, 19, 20, 21, 22, 23, 24, 25, 26, 27 및/또는 28에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 서열 190에 적어도 85%, 바람직하게는 적어도 89% 서열 동일성을 갖는 아미노산 서열을 포함하는, 보다 바람직하게는 서열 1, 19, 20, 21, 22, 23, 24, 25, 26, 27 및/또는 28에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 서열 동일성을 갖고 도 3a 내지 3d에 제시된 정렬에서 100% 보존된 모든 아미노산을 갖는 아미노산 서열을 포함하는, DmaW 활성을 갖는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하는 적어도 하나의 발현 카세트
를 포함하는 재조합 미생물. - 제5항, 제6항, 및 제8항 내지 제17항 중 어느 한 항에 있어서, ERG9 활성이 서열 9에 적어도 70% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드에 의한 것인 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체.
- 제5항 내지 제17항 중 어느 한 항에 있어서, ERG20 활성이 서열 10에 적어도 70% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드에 의한 것인 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 또는 재조합 폴리뉴클레오티드.
- 제5항, 제6항, 및 제8항 내지 제17항 중 어느 한 항에 있어서, HMG-CoA 리덕타제 활성이 서열 11에 적어도 70% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드에 의한 것인 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체.
- 제20항에 있어서, ERG9, ERG20 및 HMG-CoA 리덕타제 활성 중 적어도 2개가 서열 9, 10 또는 11에 적어도 70% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드에 의한 것인 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체.
- 제21항에 있어서, ERG9, ERG20 및 HMG-CoA 리덕타제 활성이 서열 9, 10 및 11에 적어도 70% 서열 동일성을 갖는 아미노산 서열을 포함하는 폴리펩티드에 의한 것인 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체.
- a. 시클로클라빈,
b. 페스투클라빈,
c. 아그로클라빈
d. 카노클라빈 알데히드, 및
e. 카노클라빈 I
로 이루어지는 화합물의 군으로부터 선택되는 적어도 하나의 화합물을 포함하고, 천연 맥각 알칼로이드 생산자 유기체가 아닌 재조합 미생물. - 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및 카노클라빈 I의 군으로부터 선택되는 화합물 중 적어도 하나를 동일한 조건 하에 성장할 때 비-재조합 야생형 유기체에 비해 보다 많은 양으로 포함하고/하거나 생산하는 재조합 천연 맥각 알칼로이드 생산자 유기체.
- 제1항 내지 제6항, 및 제8항 내지 제22항 중 어느 한 항에 있어서,
a. 시클로클라빈,
b. 페스투클라빈,
c. 아그로클라빈,
d. 카노클라빈 알데히드, 및
e. 카노클라빈 I
로 이루어지는 화합물의 군으로부터 선택되는 적어도 하나의 화합물을 포함하는 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체. - 제7항 내지 제16항 중 어느 한 항에서 설명된 폴리펩티드를 코딩하는 적어도 하나의 폴리뉴클레오티드의 발현을 위한 적어도 2개의 발현 카세트를 포함하는 재조합 폴리뉴클레오티드, 바람직하게는 벡터.
- a) 제1항 내지 제6항, 및 제8항 내지 제25항 중 어느 한 항에 따른 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체에서 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나의 생산을 허용하는 조건 하에 상기 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체를 배양하는 단계; 및
b) 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 및/또는 배양 배지로부터 상기 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및/또는 카노클라빈 I을 얻는 단계
를 포함하는, 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나의 생산 방법. - a) 제1항 내지 제6항, 및 제8항 내지 제25항 중 어느 한 항에 따른 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체에서 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 카노클라빈 I 중 적어도 하나의 생산을 허용하는 조건 하에 상기 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체를 배양하는 단계;
b) 상기 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체에게 IPP, 트립토판, DMAPP, DMAT, Me-DMAT 또는 카노클라빈 I의 군으로부터 선택되는 화합물 중 적어도 하나를 배양 배지를 통해 제공하는 단계; 및
c) 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체 및/또는 배양 배지로부터 상기 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및/또는 카노클라빈 I을 얻는 단계
를 포함하는, 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나의 생산 방법. - 제27항 또는 제28항에 있어서, 시클로클라빈이 생산되고 얻어지는 것인 방법.
- 제27항 내지 제29항 중 어느 한 항에 있어서, 카노클라빈 알데히드가 배지에 공급되고, 시클로클라빈이 생산되고 얻어지는 것인 방법.
- 제27항 내지 제30항 중 어느 한 항에 있어서, 카노클라빈 I이 배지에 공급되고, 시클로클라빈이 생산되고 얻어지는 것인 방법.
- 제27항 내지 제31항 중 어느 한 항에 있어서, Me-DMAT가 배지에 공급되고, 시클로클라빈이 생산되고 얻어지는 것인 방법.
- 제27항 내지 제32항 중 어느 한 항에 있어서, DMAT가 배지에 공급되고, 시클로클라빈이 생산되고 얻어지는 것인 방법.
- 제27항 내지 제33항 중 어느 한 항에 있어서, 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체가 10℃ 내지 40℃의 온도에서 배양되는 것인 방법.
- 제27항 내지 제34항 중 어느 한 항에 있어서, 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체가 10℃ 내지 32℃의 온도에서 배양되는 것인 방법.
- 제27항 내지 제35항 중 어느 한 항에 있어서, 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체가 먼저 25℃ 내지 40℃, 바람직하게는 25℃ 내지 35℃의 온도에서, 이어서 10℃ 내지 25℃, 바람직하게는 15℃ 내지 25℃의 온도에서 배양되거나, 또는 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체가 먼저 10℃ 내지 25℃, 바람직하게는 15℃ 내지 25℃의 온도에서, 이어서 25℃ 내지 40℃, 바람직하게는 25℃ 내지 35℃의 온도에서 배양되는 것인 방법.
- 제27항 내지 제36항 중 어느 한 항에 있어서, 생산된 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나가 추출을 통해 얻어지는 것인 방법.
- 제27항, 제28항 및 제37항 중 어느 한 항에 있어서, 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나가, 에틸 아세테이트 (EA), tert-부틸 메틸에테르 (TBME), 클로로포름 (CHCl3) 및 디클로로메탄 (DCM)의 군으로부터 선택된 화합물을 사용한 추출을 포함하는 추출 방법을 통해 얻어지는 것인 방법.
- 제27항, 제28항, 제37항 및 제38항 중 어느 한 항에 있어서, 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나가 pH=10에서 에틸 아세테이트를 사용한 액체/액체 추출, 바람직하게는 pH가 NaOH로 조정되는, pH=10에서 에틸 아세테이트를 사용한 액체/액체 추출을 포함하는 추출 방법을 통해 얻어지는 것인 방법.
- 제27항, 제28항, 및 제37항 내지 제39항 중 어느 한 항에 있어서, 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나가 바람직하게는 pH=3의 HLB 수지, pH=3의 디아이온(Diaion) 수지, pH=3의 앰벌라이트(Amberlite) 1180 수지, pH=10의 앰벌라이트 1180 수지, pH=3의 앰벌라이트 16N 수지 또는 pH=10의 앰벌라이트 16N 수지의 군으로부터 선택되는 실리카계 수지 물질을 사용하는 고상 추출을 이용한 추출을 포함하는 추출 방법을 통해 얻어지는 것인 방법.
- 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나의 생산을 위한, 제1항 내지 제6항 중 어느 한 항, 또는 제8항 내지 제25항 중 어느 한 항에 따른 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체, 또는 제7항 내지 제16항 중 어느 한 항에 따른 재조합 폴리뉴클레오티드, 또는 제26항에 따른 벡터의 용도.
- 제27항 내지 제40항 중 어느 한 항에 따른 방법에 있어서, 제1항 내지 제6항 중 어느 한 항, 또는 제8항 내지 제25항 중 어느 한 항에 따른 재조합 미생물 또는 재조합 천연 맥각 알칼로이드 생산자 유기체, 또는 제7항 내지 제16항 중 어느 한 항에 따른 재조합 폴리뉴클레오티드, 또는 제26항에 따른 벡터의 용도.
- 제27항 내지 제40항 중 어느 한 항에 따른 방법에 의해 생산된 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I.
- 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나를 포함하지만, 다른 맥각 알칼로이드는 존재하지 않거나 낮은 함량으로 존재하는, 제27항 내지 제40항 중 어느 한 항에 따른 방법에서 생산된 배양 배지.
- 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나를 포함하지만, 다른 맥각 알칼로이드는 존재하지 않거나 낮은 함량으로 존재하는 배양 배지.
- a) 서열 129, 130, 131, 132, 133, 134, 135 또는 136에 제시된 뉴클레오티드 서열을 갖는 핵산 서열;
b) 서열 20, 31, 41, 53, 64, 75, 86, 95 또는 180에 제시된 아미노산 서열을 갖는 폴리펩티드를 코딩하는 핵산 서열;
c) 서열 129, 130, 131, 132, 133, 134, 135 또는 136에 제시된 핵산 서열에 적어도 70% 동일하고 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 리덕타제, EasA 이소머라제 또는 EasG 활성을 갖는 폴리펩티드를 코딩하는 핵산 서열;
d) DmaW, EasF, EasE, EasC, EasD, EasH, EasA 리덕타제, EasA 이소머라제 또는 EasG 활성을 갖고 서열 20, 31, 41, 53, 64, 75, 86 또는 180에 제시된 아미노산 서열에 적어도 70% 동일한 아미노산 서열을 갖는 폴리펩티드를 코딩하는 핵산 서열; 및
e) 엄격한 조건 하에 a) 내지 d) 중 임의의 하나에 혼성화할 수 있고 DmaW, EasF, EasE, EasC, EasD, EasH, EasA 리덕타제, EasA 이소머라제 또는 EasG 활성을 갖는 폴리펩티드를 코딩하는 핵산 서열
로 이루어지는 군으로부터 선택되는 하나 이상의 핵산 서열을 포함하는 폴리뉴클레오티드. - a) 서열 175, 176, 177 또는 178에 제시된 아미노산 서열을 갖는 폴리펩티드를 코딩하는 핵산 서열;
b) EasE 활성을 갖고 서열 175, 176, 177 또는 178에 제시된 아미노산 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 동일한 아미노산 서열을 갖는 폴리펩티드를 코딩하는 핵산 서열;
c) 서열 181에 제시된 아미노산 서열을 갖는 폴리펩티드를 코딩하는 핵산 서열, 및
d) DmaW 활성을 갖고 서열 181에 제시된 아미노산 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 동일한 아미노산 서열을 갖는 폴리펩티드를 코딩하는 핵산 서열
로 이루어지는 군으로부터 선택된 하나 이상의 핵산 서열을 포함하는 폴리뉴클레오티드. - 제46항 또는 제47항에 있어서, 상기 폴리뉴클레오티드가 제8항 내지 제17항 중 어느 한 항에 규정된 폴리펩티드를 코딩하는 핵산 서열을 추가로 포함하는 것인 폴리뉴클레오티드.
- 제46항 또는 제47항의 폴리뉴클레오티드를 포함하는, 또는 제48항의 폴리뉴클레오티드를 포함하는 발현 카세트를 포함하는 벡터 또는 발현 카세트.
- 제49항에 있어서, 발현 벡터인 벡터.
- 제46항 내지 제48항 중 어느 한 항에 따른 폴리뉴클레오티드 또는 제49항 또는 제50항에 따른 벡터를 포함하는 숙주 세포.
- 제51항에 있어서, 박테리아 세포, 진균 세포, 또는 효모 세포인 숙주 세포.
- 제51항 또는 제52항에 있어서, 박테리아 세포, 진균 세포, 또는 효모 세포, 바람직하게는 아스페르길루스 자포니쿠스(Aspergillus japonicus), 아스페르길루스 푸미가투스(Aspergillus fumigatus), 클라비셉스 푸르푸레아(Claviceps purpurea), 클라비셉스 파스팔리(Claviceps paspali), 클라비셉스 아프리카누스(Claviceps africanus) 또는 효모 세포인 숙주 세포.
- DmaW, EasF, EasE, EasC, EasD, EasH, EasA 리덕타제, EasA 이소머라제 또는 EasG 활성을 갖고 서열 20, 31, 41, 53, 64, 75, 86, 또는 180에 제시된 아미노산 서열에 적어도 70% 동일한 아미노산 서열을 갖는 폴리펩티드를 과다발현하고/하거나
EasE 활성을 갖고 서열 175, 176, 177 또는 178에 제시된 아미노산 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 동일한 아미노산 서열을 갖는 폴리펩티드 및/또는 DmaW 활성을 갖고 서열 181에 제시된 아미노산 서열에 적어도 70%, 72%, 74%, 76%, 78%, 80%, 82%, 84%, 86%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 또는 100% 동일한 아미노산 서열을 갖는 폴리펩티드를 과다발현하는 파에실로미세스 디바리카투스(Paecilomyces divaricatus). - a) 제51항 내지 제53항 중 어느 한 항에 따른 숙주 세포 또는 파에실로미세스 디바리카투스 세포를 제46항 또는 제47항에 따른 폴리뉴클레오티드에 의해 코딩되는 폴리펩티드의 생산을 허용하는 조건 하에 배양하는 단계; 및
b) 단계 a)의 숙주 세포 또는 파에실로미세스 디바리카투스로부터 폴리펩티드를 얻는 단계
를 포함하는, 제46항 또는 제47항에 따른 폴리뉴클레오티드에 의해 코딩되는 적어도 하나의 폴리펩티드의 제조 방법. - 제46항 또는 제47항의 폴리뉴클레오티드에 의해 코딩되거나 또는 제55항의 방법에 의해 얻을 수 있는 폴리펩티드.
- 제56항의 폴리펩티드에 특이적으로 결합하는 항체.
- a) 제51항 내지 제53항 중 어느 한 항에 따른 숙주 세포 또는 파에실로미세스 디바리카투스 또는 제54항에 따른 파에실로미세스 디바리카투스를, 상기 숙주 세포에서 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나의 생산을 허용하는 조건 하에서 배양하는 단계; 및
b) 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I 중 적어도 하나를 상기 숙주 세포 또는 파에실로미세스 디바리카투스 세포 또는 배양 배지로부터 얻는 딘계
를 포함하는, 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 또는 카노클라빈 I, 또는 이들의 조합물의 제조 방법. - 시클로클라빈, 페스투클라빈, 아그로클라빈, 카노클라빈 알데히드 및 카노클라빈 I의 군으로부터 선택되는 화합물 중 적어도 하나의 제조를 위한, 제46항 또는 제47항에 따른 폴리뉴클레오티드, 제49항 또는 제50항에 따른 벡터 또는 제51항 내지 제53항 중 어느 한 항에 따른 숙주 세포 또는 파에실로미세스 디바리카투스의 용도.
Applications Claiming Priority (11)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201261691848P | 2012-08-22 | 2012-08-22 | |
| EP12181388 | 2012-08-22 | ||
| EP12181388.5 | 2012-08-22 | ||
| US61/691,848 | 2012-08-22 | ||
| US201361786841P | 2013-03-15 | 2013-03-15 | |
| EP13159444 | 2013-03-15 | ||
| US61/786,841 | 2013-03-15 | ||
| EP13159444.2 | 2013-03-15 | ||
| EP13178008 | 2013-07-25 | ||
| EP13178008.2 | 2013-07-25 | ||
| PCT/IB2013/056604 WO2014030096A2 (en) | 2012-08-22 | 2013-08-13 | Genes and processes for the production of clavine-type alkaloids |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20150042856A true KR20150042856A (ko) | 2015-04-21 |
Family
ID=50150439
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020157006881A KR20150042856A (ko) | 2012-08-22 | 2013-08-13 | 클라빈-유형 알칼로이드의 생산을 위한 유전자 및 방법 |
Country Status (12)
| Country | Link |
|---|---|
| US (1) | US9828617B2 (ko) |
| EP (1) | EP2888351A4 (ko) |
| JP (1) | JP2015530879A (ko) |
| KR (1) | KR20150042856A (ko) |
| CN (1) | CN104736696B (ko) |
| AU (1) | AU2013307289A1 (ko) |
| BR (1) | BR112015003695A8 (ko) |
| CA (1) | CA2880845A1 (ko) |
| IL (1) | IL237170A0 (ko) |
| IN (1) | IN2015DN00922A (ko) |
| MX (1) | MX2015002294A (ko) |
| WO (1) | WO2014030096A2 (ko) |
Families Citing this family (9)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| KR20210133323A (ko) | 2013-12-13 | 2021-11-05 | 바스프 에스이 | 정밀 화학물질의 개선된 생산을 위한 재조합 미생물 |
| US9790528B2 (en) * | 2014-06-16 | 2017-10-17 | West Virginia University | Production of lysergic acid by genetic modification of a fungus |
| CN107454915B (zh) | 2015-03-26 | 2022-03-22 | 巴斯夫欧洲公司 | L-岩藻糖的生物催化生产方法 |
| US20210002678A1 (en) * | 2018-03-20 | 2021-01-07 | Manus Bio, Inc. | Enzymes, cells and methods for production of 3-(4-farnesyloxyphenyl)propionic acid and derivatives thereof |
| EP3714959A1 (en) * | 2019-03-29 | 2020-09-30 | Basf Se | Isolation of small molecules from the fermentation broth of a eukaryotic microorganism |
| CZ308933B6 (cs) * | 2019-07-19 | 2021-09-15 | Univerzita Palackého v Olomouci | Houba Claviceps purpurea se zvýšenou produkcí námelových alkaloidů, způsob její přípravy, expresní kazeta |
| CN113308442B (zh) * | 2021-04-28 | 2023-06-27 | 天津大学 | 重组酿酒酵母菌株及其构建方法 |
| CN113174399A (zh) * | 2021-04-28 | 2021-07-27 | 天津大学 | 重组酿酒酵母菌株及其发酵方法 |
| CN116103351A (zh) * | 2023-03-06 | 2023-05-12 | 天津大学 | 高产田麦角碱重组酵母构建方法与应用 |
Family Cites Families (4)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US7192751B2 (en) | 2001-12-06 | 2007-03-20 | The Regents Of The University Of California | Biosynthesis of amorpha-4,11-diene |
| US7172886B2 (en) | 2001-12-06 | 2007-02-06 | The Regents Of The University Of California | Biosynthesis of isopentenyl pyrophosphate |
| DE06828944T1 (de) | 2005-11-07 | 2008-01-10 | Ivax Pharmaceuticals S.R.O. | PILZSTAMM CLAVICEPS PURPUREA FÜR DIE INDUSTRIELLE PRODUKTION (Fr.) Tul. |
| AU2012222492A1 (en) | 2011-03-03 | 2013-08-29 | Basf Se | Gene cluster for biosynthesis of cycloclavine |
-
2013
- 2013-08-13 JP JP2015527987A patent/JP2015530879A/ja active Pending
- 2013-08-13 CA CA2880845A patent/CA2880845A1/en active Pending
- 2013-08-13 WO PCT/IB2013/056604 patent/WO2014030096A2/en active Application Filing
- 2013-08-13 BR BR112015003695A patent/BR112015003695A8/pt not_active IP Right Cessation
- 2013-08-13 MX MX2015002294A patent/MX2015002294A/es unknown
- 2013-08-13 CN CN201380054217.4A patent/CN104736696B/zh not_active Expired - Fee Related
- 2013-08-13 US US14/421,872 patent/US9828617B2/en not_active Expired - Fee Related
- 2013-08-13 IN IN922DEN2015 patent/IN2015DN00922A/en unknown
- 2013-08-13 KR KR1020157006881A patent/KR20150042856A/ko not_active Application Discontinuation
- 2013-08-13 EP EP13831449.7A patent/EP2888351A4/en not_active Withdrawn
- 2013-08-13 AU AU2013307289A patent/AU2013307289A1/en not_active Abandoned
-
2015
- 2015-02-10 IL IL237170A patent/IL237170A0/en unknown
Also Published As
| Publication number | Publication date |
|---|---|
| IL237170A0 (en) | 2015-04-30 |
| AU2013307289A1 (en) | 2015-02-19 |
| US9828617B2 (en) | 2017-11-28 |
| IN2015DN00922A (ko) | 2015-06-12 |
| WO2014030096A2 (en) | 2014-02-27 |
| JP2015530879A (ja) | 2015-10-29 |
| CN104736696A (zh) | 2015-06-24 |
| MX2015002294A (es) | 2015-10-09 |
| CN104736696B (zh) | 2018-08-28 |
| CA2880845A1 (en) | 2014-02-27 |
| BR112015003695A2 (pt) | 2017-12-12 |
| BR112015003695A8 (pt) | 2018-04-10 |
| EP2888351A4 (en) | 2016-04-27 |
| WO2014030096A3 (en) | 2014-04-17 |
| US20150211036A1 (en) | 2015-07-30 |
| EP2888351A2 (en) | 2015-07-01 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| CN108026523B (zh) | 向导rna组装载体 | |
| KR20150042856A (ko) | 클라빈-유형 알칼로이드의 생산을 위한 유전자 및 방법 | |
| CN101939434B (zh) | 用于在大豆中提高种子贮藏油脂的生成和改变脂肪酸谱的来自解脂耶氏酵母的dgat基因 | |
| CA2774975C (en) | Production of fatty acid derivatives in recombinant bacterial cells expressing an ester synthase variant | |
| AU609783B2 (en) | Novel fusion proteins and their purification | |
| CN107250163A (zh) | 修饰的葡糖淀粉酶和具有增强的生物产物产生的酵母菌株 | |
| DK3027733T3 (en) | Preparation of 3-Hydroxypropionic Acid in Recombinant Yeast Expressing an Insect Aspartate-1 Decarboxylase | |
| CN108138121B (zh) | 用微生物高水平生产长链二羧酸 | |
| CN101889088B (zh) | 从植物基因组中切除核酸序列的方法 | |
| IL236992A (en) | Genetically modified cyanobacteria that produce ethanol | |
| DK3108014T3 (en) | Methods and systems for rapid detection of microorganisms using recombinant bacteriophage | |
| KR20130027063A (ko) | Fe-s 클러스터 요구성 단백질의 활성 향상 | |
| KR20140113997A (ko) | 부탄올 생성을 위한 유전자 스위치 | |
| KR20130117753A (ko) | 포스포케톨라아제를 포함하는 재조합 숙주 세포 | |
| CN108495685B (zh) | 针对艰难梭菌感染的基于酵母的免疫疗法 | |
| KR20070085669A (ko) | 고농도의 아라키돈산을 생성하는 야로위아 리폴리티카 균주 | |
| CN111763686B (zh) | 实现c到a以及c到g碱基突变的碱基编辑系统及其应用 | |
| CN108753778B (zh) | 利用碱基编辑修复fbn1t7498c突变的试剂和方法 | |
| CN108431229A (zh) | 糖转运蛋白修饰的酵母菌株和用于生物制品生产的方法 | |
| KR102303832B1 (ko) | 내산성을 갖는 효모 세포, 상기 효모 세포를 제조하는 방법 및 이의 용도 | |
| CN108368490A (zh) | 真菌产生fdca | |
| CN114181957B (zh) | 一种基于病毒加帽酶的稳定t7表达系统及其在真核生物中表达蛋白质的方法 | |
| KR20180084135A (ko) | 감소된 clr2 활성을 갖는 사상 진균에서 단백질을 생산하는 방법 | |
| CN111088176B (zh) | 产β-胡萝卜素的基因工程菌及其应用 | |
| KR102311681B1 (ko) | 내산성을 갖는 효모 세포, 그를 이용하여 유기산을 생산하는 방법 및 상기 내산성 효모 세포를 생산하는 방법 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| WITN | Application deemed withdrawn, e.g. because no request for examination was filed or no examination fee was paid |