KR20120089631A - 개선된 당 수송, 혼합된 당 발효 및 바이오 연료의 생산을 위한 방법 및 조성물 - Google Patents
개선된 당 수송, 혼합된 당 발효 및 바이오 연료의 생산을 위한 방법 및 조성물 Download PDFInfo
- Publication number
- KR20120089631A KR20120089631A KR1020127002889A KR20127002889A KR20120089631A KR 20120089631 A KR20120089631 A KR 20120089631A KR 1020127002889 A KR1020127002889 A KR 1020127002889A KR 20127002889 A KR20127002889 A KR 20127002889A KR 20120089631 A KR20120089631 A KR 20120089631A
- Authority
- KR
- South Korea
- Prior art keywords
- helix
- host cell
- cells
- polypeptide
- transmembrane
- Prior art date
Links
- 0 C([C@@]12)=C3C=C*4=C1[C@]24C=C3 Chemical compound C([C@@]12)=C3C=C*4=C1[C@]24C=C3 0.000 description 2
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P21/00—Preparation of peptides or proteins
- C12P21/02—Preparation of peptides or proteins having a known sequence of two or more amino acids, e.g. glutathione
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P7/00—Preparation of oxygen-containing organic compounds
- C12P7/02—Preparation of oxygen-containing organic compounds containing a hydroxy group
- C12P7/04—Preparation of oxygen-containing organic compounds containing a hydroxy group acyclic
- C12P7/06—Ethanol, i.e. non-beverage
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/37—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from fungi
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/705—Receptors; Cell surface antigens; Cell surface determinants
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N1/00—Microorganisms, e.g. protozoa; Compositions thereof; Processes of propagating, maintaining or preserving microorganisms or compositions thereof; Processes of preparing or isolating a composition containing a microorganism; Culture media therefor
- C12N1/14—Fungi; Culture media therefor
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/52—Genes encoding for enzymes or proenzymes
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/80—Vectors or expression systems specially adapted for eukaryotic hosts for fungi
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/80—Vectors or expression systems specially adapted for eukaryotic hosts for fungi
- C12N15/81—Vectors or expression systems specially adapted for eukaryotic hosts for fungi for yeasts
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/87—Introduction of foreign genetic material using processes not otherwise provided for, e.g. co-transformation
-
- Y—GENERAL TAGGING OF NEW TECHNOLOGICAL DEVELOPMENTS; GENERAL TAGGING OF CROSS-SECTIONAL TECHNOLOGIES SPANNING OVER SEVERAL SECTIONS OF THE IPC; TECHNICAL SUBJECTS COVERED BY FORMER USPC CROSS-REFERENCE ART COLLECTIONS [XRACs] AND DIGESTS
- Y02—TECHNOLOGIES OR APPLICATIONS FOR MITIGATION OR ADAPTATION AGAINST CLIMATE CHANGE
- Y02E—REDUCTION OF GREENHOUSE GAS [GHG] EMISSIONS, RELATED TO ENERGY GENERATION, TRANSMISSION OR DISTRIBUTION
- Y02E50/00—Technologies for the production of fuel of non-fossil origin
- Y02E50/10—Biofuels, e.g. bio-diesel
Landscapes
- Life Sciences & Earth Sciences (AREA)
- Health & Medical Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Genetics & Genomics (AREA)
- Organic Chemistry (AREA)
- Engineering & Computer Science (AREA)
- Zoology (AREA)
- Wood Science & Technology (AREA)
- Biotechnology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- General Engineering & Computer Science (AREA)
- Biomedical Technology (AREA)
- Biochemistry (AREA)
- General Health & Medical Sciences (AREA)
- Molecular Biology (AREA)
- Microbiology (AREA)
- Biophysics (AREA)
- Mycology (AREA)
- Plant Pathology (AREA)
- Physics & Mathematics (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Medicinal Chemistry (AREA)
- Gastroenterology & Hepatology (AREA)
- Chemical Kinetics & Catalysis (AREA)
- General Chemical & Material Sciences (AREA)
- Cell Biology (AREA)
- Toxicology (AREA)
- Immunology (AREA)
- Botany (AREA)
- Virology (AREA)
- Tropical Medicine & Parasitology (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Preparation Of Compounds By Using Micro-Organisms (AREA)
- Enzymes And Modification Thereof (AREA)
- Peptides Or Proteins (AREA)
Abstract
본 발명은 세포 내로 셀로덱스트린을 수송하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포에 관한 것이다. 본 발명은 추가로 세포 내로 셀로덱스트린의 수송을 증가시키는 방법, 셀로덱스트린을 포함하는 배지에서 세포의 성장을 증가시키는 방법, 셀룰로스-유도 및 헤미셀룰로스-유도 당을 공동-발효시키는 방법, 및 세포 내로 셀로덱스트린을 수송하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 준비하여 탄화수소 또는 탄화수소 유도체를 제조하는 방법에 관한 것이다. 본 발명은 세포 내로 오탄당을 수송하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포, 세포 내로 오탄당의 수송을 증가시키는 방법, 오탄당 당을 포함하는 배지에서 세포의 성장을 증가시키는 방법 및 세포 내로 오탄당을 수송하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 준비하여 탄화수소 또는 탄화수소 유도체를 제조하는 방법에 관한 것이다.
Description
관련된 출원서(
RELATED
APPLICATIONS
)에 대한 참고문헌(
CROSS
-
REFERENCE
)
본 출원서는, 2009년 12월 10일 미국 가출원 번호 61/285,526 및 2009년 7월 24일 출원된 미국 가출원 번호 61/271,833 의 내용을 청구하고, 이들 모두는 전체가 본원의 참고문헌으로 인용된다.
본 발명의 분야
본 발명은, 세포 내에 당의 수송(transport)을 증가시키기 위한, 세포의 성장을 증가시키기 위한, 탄화수소 및 탄화수소 유도체(hydrocarbon derivatives)의 합성을 증가시키기 위한, 셀룰로스-유도 당(cellulose-derived sugars) 및 헤미셀룰로스-유도 당(hemicellulose-derived sugars)을 공동 발효시키기 위한(co-fermenting) 방법 및 조성물에 관한 것이다.
바이오 연료(biofuels)는, 에너지 안전 보장(energy security), 지속가능성(sustainablility), 및 지구 기후 변화(global climate change)에 대한 증가된 우려(concerns)때문에 집중적인 조사 하에 있다(Lynd 등의 1991). 식물-유도 리그노셀룰로스계 물질(plant-derived lignocellulosic material)을 바이오 연료로의 생물학적 변환(bioconversion)은 화석연료(fossil fuel)의 화학적인 생산에 대한 흥미로운 대안으로서 간주되고 있다(Lynd 등의 2008 ; Hahn-Hagerdal 등의 2006). 리그노셀룰로스 바이오매스(lignocellulose biomass)는 셀룰로스(cellulose), 헤미셀룰로스(hemicellulose) 및 리그닌(lignin)으로 구성된다.
리그노셀룰로스계 바이오매스를 에탄올로의 전환을 유효하게 실행하기 위해 미생물 공학은 바이오 연료 분야(biofuels field)의 주요한 목표로 여전히 남아있다. 대부분의 조사는, 셀룰라아제(cellulases) 및 그 밖의 효소의 발현에 의해 단순한 당(sugars)이 알코올로 자연적으로 발효되는 유전적으로 조작된 미생물에 집중되어 있고, 이들은 리그노셀룰로스계 바이오매스 중합체(lignocellulosic biomass polymers)를 분해하여 하나의 세포 내에서 에탄올을 발생시킨다. 그러나, 연구가 잘 되지 않는 영역은 당 운반체(sugar transport) 영역이다. 당-흡수 능력(sugar-uptake ability)을 개선시키기 위한 당 운반체의 조절 및 미생물의 유전공학의 이해는 매우 효율을 증진시킬 것이다(Stephanopoulos 2007). 더욱이, 셀룰라아제 발현 및 활성의 조절에서 포함되는 그 밖의 유형의 단백질은 연구할 부분이 많이 남아있다.
빵 효모(baker's yeast)로 또한 알려진 사카로미세스 세레비시아(Saccharomyces cerevisiae)는 수 천년 동안 6 탄당(hexose sugars)을 에탄올로 생물전환(bioconversion)에 사용된다. 이는 또한 D-글루코스에서 에탄올로의 대규모 발효 공업에 대해서 미생물을 매우 넓게 사용되고 있다. 에스 . 세레비시아(S. cerevisiae)는 리그노셀룰로스계 바이오매스를 바이오연료(biofuel)로의 생물전환의 매우 적절한 후보물질이다(van Maris 등의, 2006). 이는 유전적이고 생리학적인 배경기술, 충분한 유전적 도구 및 리그노셀룰로스계 가수분해(lignocellulosic hydrolysates)에서 나타낸 높은 에탄올 농도 및 저해제에 대한 높은 내성(tolerance)에 대해서는 잘 연구되어 있다(Jeffries 2006). 에스 . 세레 비시아의 낮은 발효 pH 는 발효하는 동안에 세균 오염(bacterial contamination)에 또한 제시될 수 있다.
유감스럽게도, 야생형 에스 . 세레비시아는 오탄당 당(pentose sugars)에서 사용할 수 없다(Hector 등의, 2008). 이러한 한계를 극복하기 위해, 오탄당-소화하는 유기체(pentose-assimilating organisms)를 에스 . 세레비시아에 도입하였고, 이는 D-자일로스(D-xylose) 및 L-아라비노스(L-arabinose)의 발효를 가능하게 한다(Hahn-Hagerdal 등의, 2007; Brat 등의, 2009; Wisselink 등의, 2007, 2009; Wiedemann and Boles 2008; Karhumma 등의, 2006). 그러나, 오탄당을 바이오연료로의 효율적인 전환은, 세포내 산화환원 반응의 불균형(cellular redox imbalance), 5 탄당 인산경료(pentose phosphate pathway)의 낮은 유입(low influx) 및 세포 내로의 효율적인 5 탄당 운송의 결핍을 포함하는 다양한 문제에 의해 제한되고 있다(Hector 등의, 2008).
게다가, 자연적인 미생물 및 유전자 조작된 미생물 모두는, 글루코스 발표와 비교하여 자일로스 발효하는 동안에 감소된 에탄올 내성(ethanol tolerance)을 나타내었다(Jeffries and Jin 2000). 더 낮은 발효 비율과 결합하여, 자일로스 발효 동안의 감소된 에탄올 내성은 셀룰로스 가수분해물(cellulosic hydrolysates)에 있는 높은 농도의 글루코스(~ 70 - 100 g/L) 및 자일로스(~ 40 - 60 g/L)를 함유하는 당 혼합물의 발효에서 중요한 문제로 제시되고 있다. 미생물이 우선적으로 글루코스를 사용하기 때문에, 글루코스가 고갈된 시기(세포가 자일로스를 사용하기 시작할 때)에, 에탄올 농도는 자일로스 발효 비율을 추가적으로 감소시키기에 이미 충분히 높다(~ 35-45 g/L). 결과적으로, "글루코스의 억압(glucose repression)" 때문에 글루코스가 고갈된 후에의 자일로스의 순차적인 이용은, 셀룰로스 가수분해물(cellulosic hydrolysates)에서 혼합된 당을 성공적으로 사용하기 위하여 극복하기 위한 중요한 도전이다.
따라서, 리그노셀룰로스의 분해에 중요한 부가적인 유전자의 식별, 및 리그노셀룰로스에서의 개선된 성장을 위한 미생물의 공학(engineering)에서의 이들의 사용 및 리그노셀룰로스 분해의 결과로 초래된 화합물의 활용을 위한 필요성이 존재한다. 또한 5 탄당에서 바이오연료로의 효과적인 전환을 위한 개선된 방법 및 바이오연료를 생산하기 위한 혼합된 당의 발효를 위한 개선된 방법이 추가적으로 필요하다.
본 발명의 간단한 요약
이러한 필요성을 충족시키기 위해, 본원에 기재된 본 발명은 세포 내로의 셀로덱스트린의 수송(transport)을 증가시키는 방법, 셀로덱스트린을 함유하는 배지에서 세포의 성장을 증가시키는 방법, 셀룰로스-유도(cellulose-derived) 및 헤미셀룰로스-유도 당(hemicellulose-derived sugars)을 공동-발효시키는(co-fermenting) 방법 및 세포 내로 셀로덱스트린을 수송하는 폴리펩티드를 코딩하는(encoding) 재조합 폴리뉴클레오티드(recombinant polynucleotide)를 함유하는 숙주 세포(host cell)를 제공하여 탄화수소(hydrocarbons) 또는 탄화수소 유도체(hydrocarbon derivatives)를 생성하는 방법을 제공하고자 한다. 추가적으로 기재된 숙주 세포는, 세포 내로 셀로덱스트린을 수송하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 함유한다. 추가적으로 본 발명은, 세포 내로 오탄당을 수송하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 함유하는 숙주 세포, 세포 내로 오탄당의 수송을 증가시키는 방법, 오탄당을 함유하는 배지에서 세포의 성장을 증가시키는 방법, 및 세포 내로 오탄당을 수송하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 함유하는 숙주 세포를 제공하여 탄화수소 또는 탄화수소 유도체를 제조하는 방법에 관한 것이다.
본원에서 사용된 바와 같이, 셀로덱스트린은, 셀로비오스(cellobiose)(2 글루코스 단량체), 셀로트로스(cellotriose)(3 글루코스 단량체), 셀로테트라오스(cellotetraose)(4 글루코스 단량체), 셀로펜타오스(cellopentaose)(5 글루코스 단량체) 및 셀로헥사오스(cellohexaose)(6 글루코스 단량체)를 포함하는, 다양한 길이(length)의 글루코스 중합체에 관한 것이다.
따라서, 한 양상에 있어서, 트랜스멤브레인(transmembrane) α-헬릭스 1(α-helix 1), α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드(polypeptide)를 코딩하는(encoding) 재조합 폴리뉴클레오티드(recombinant polynucleotide)를 포함하는, 상기 트랜스멤브레인 α-헬릭스 1 은 SEQ ID NO : 1 을 포함하는 숙주 세포를 준비하고, 재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로의 셀로덱스트린의 수송이 증가되는, 재조합 폴리뉴클레오티드가 발현되는 배지(medium)에서 세포를 배양하는 것을 포함하는, 세포 내로 셀로덱스트린 수송을 증가시키는 방법을 포함한다.
또 다른 양상에 있어서, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는, 상기 트랜스멤브레인 α-헬릭스 2 는 SEQ ID NO : 2 을 포함하는 숙주 세포를 준비하고, 재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로의 셀로덱스트린의 수송이 증가되는, 재조합 폴리뉴클레오티드가 발현되는 배지(medium)에서 세포를 배양하는 것을 포함하는, 세포 내로 셀로덱스트린 수송을 증가시키는 방법을 포함한다.
또 다른 양상에 있어서, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는, 상기 트랜스멤브레인 α-헬릭스 2 및 트랜스멤브레인 α-헬릭스 3 을 연결하는 루프(loop)는 SEQ ID NO : 3 을 포함하는 숙주 세포를 준비하고, 재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로의 셀로덱스트린의 수송이 증가되는, 재조합 폴리뉴클레오티드가 발현되는 배지(medium)에서 세포를 배양하는 것을 포함하는, 세포 내로 셀로덱스트린 수송을 증가시키는 방법을 포함한다.
또 다른 양상에 있어서, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는, 상기 트랜스멤브레인 α-헬릭스 5 는 SEQ ID NO : 4 을 포함하는 숙주 세포를 준비하고, 재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로의 셀로덱스트린의 수송이 증가되는, 재조합 폴리뉴클레오티드가 발현되는 배지(medium)에서 세포를 배양하는 것을 포함하는, 세포 내로 셀로덱스트린 수송을 증가시키는 방법을 포함한다.
또 다른 양상에 있어서, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는, 상기 트랜스멤브레인 α-헬릭스 6 은 SEQ ID NO : 5 을 포함하는 숙주 세포를 준비하고, 재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로의 셀로덱스트린의 수송이 증가되는, 재조합 폴리뉴클레오티드가 발현되는 배지(medium)에서 세포를 배양하는 것을 포함하는, 세포 내로 셀로덱스트린 수송을 증가시키는 방법을 포함한다.
또 다른 양상에 있어서, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는, 상기 트랜스멤브레인 α-헬릭스 6 및 상기 트랜스멤브레인 α-헬릭스 7 사이의 서열은 SEQ ID NO : 6 을 포함하는 숙주 세포를 준비하고, 재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로의 셀로덱스트린의 수송이 증가되는, 재조합 폴리뉴클레오티드가 발현되는 배지(medium)에서 세포를 배양하는 것을 포함하는, 세포 내로 셀로덱스트린 수송을 증가시키는 방법을 포함한다.
또 다른 양상에 있어서, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는, 상기 트랜스멤브레인 α-헬릭스 7 은 SEQ ID NO : 7 을 포함하는 숙주 세포를 준비하고, 재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로의 셀로덱스트린의 수송이 증가되는, 재조합 폴리뉴클레오티드가 발현되는 배지에서 세포를 배양하는 것을 포함하는, 세포 내로 셀로덱스트린 수송을 증가시키는 방법을 포함한다.
또 다른 양상에 있어서, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는, 상기 트랜스멤브레인 α-헬릭스 10, 상기 트랜스멤브레인 α-헬릭스 11 및 이들 사이의 서열은 SEQ ID NO : 8 을 포함하는 숙주 세포를 준비하고, 재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로의 셀로덱스트린의 수송이 증가되는, 재조합 폴리뉴클레오티드가 발현되는 배지에서 세포를 배양하는 것을 포함하는, 세포 내로 셀로덱스트린 수송을 증가시키는 방법을 포함한다.
전술한 양상 중 어떠한 것과 결합할 수 있는 특정 실시형태에 있어서, 상기 폴리펩티드는 NCU00801 또는 NCU08114 와 적어도 29 %, 적어도 30 %, 적어도 35 %, 적어도 40 %, 적어도 45 %, 적어도 50 %, 적어도 55 %, 적어도 60 %, 적어도 65 %, 적어도 70 %, 적어도 75 %, 적어도 80 %, 적어도 85 %, 적어도 90 %, 적어도 95 %, 적어도 99 % 또는 적어도 100 % 동일한 아미노산을 갖는다. 전술한 실시형태 중 어떠한 것과 결합할 수 있는 특정 실시형태에 있어서, 숙주 세포는 β-글루코시다아제(β-glucosidase)의 적어도 촉매 도메인(catalytic domain)을 코드하는(encodes) 두 번째 재조합 폴리뉴클레오티드를 포함한다. β-글루코시다아제의 촉매 도메인을 코딩하는 두 번째 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 갖는 전술한 실시형태와 결합할 수 있는 특정 실시형태에 있어서, β-글루코시다아제는 네우로스포라 크라사(Neurospora crassa)로부터 온 것이다. 네우로스포라 크라사로부터의 β-글루코시다아제의 적어도 촉매 도메인을 코딩하는 두 번째 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 갖는 전술한 실시형태와 결합할 수 있는 특정 실시형태에 있어서, β-글루코시다아제는 NCU00130 에 의해 코드되었다. 전술한 실시형태 중 어떠한 것과 결합할 수 있는 특정 실시형태에 있어서, 숙주 세포는 하나 또는 그 이상의 재조합 폴리뉴클레오티드를 더 포함하고, 상기 하나 또는 그 이상의 폴리뉴클레오티드는 오탄당 이용(utilization)에 포함되는 하나 또는 그 이상의 효소를 코드한다. 오탄당 이용에 포함되는 하나 또는 그 이상의 효소를 코드하는 하나 또는 그 이상의 재조합 폴리뉴클레오티드를 더 함유하는 숙주 세포를 갖는 전술한 실시형태와 결합할 수 있는 특정 실시형태에 있어서, 하나 또는 그 이상의 효소는 L-아라비노스 이성질화효소(L-arabinose isomerase), L-리블로인산화효소(L- ribulokinase), L-리블로스-5-P 4 에피머화효소(L-ribulose-5-P 4 epimerase), 자일로스 이성질화효소(xylose isomerase), 자일루로키나아제(xylulokinase), 알도스 환원효소(aldose reductase), L-아라비니톨 4-탈수소효소(L-arabinitol 4- dehydrogenase), L-자일루로스 환원효소(L- xylulose reductase) 및 자일리톨 탈수소효소로 이루어진 하나 또는 그 이상의 군으로부터 선택된 것이다. 전술한 실시형태 중 어떠한 것과 결합할 수 있는 특정 실시형태에 있어서, 숙주 세포는 오탄당 운반체를 코드하는 세 번째 재조합 폴리뉴클레오티드를 더 포함한다. 오탄당 운반체를 코드하는 세 번째 재조합 폴리뉴클레오티드를 더 포함하는 숙주 세포를 갖는 전술한 실시형태와 결합할 수 있는 특정 실시형태에 있어서, 오탄당 운반체는 NCU00821, NCU04963, NCU06138, STL12/XUT6, SUT2, SUT3, XUT1 및 XUT3 으로 이루어진 군으로부터 선택된다.
또 다른 양상에 있어서, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 상기 트랜스멤브레인 α-헬릭스 1 은 SEQ ID NO : 1 을 포함하며, 상기 폴리펩티드는 셀로덱스트린 운반체인 숙주 세포를 준비하고, 숙주 세포가 재조합 폴리뉴클레오티드를 포함하지 않는 세포 보다, 배지에서 더 빠른 비율로 성장하는, 셀로덱스트린을 함유하는 배지에서 숙주 세포를 배양하는 것을 포함하는, 세포의 성장을 증가시키는 방법을 포함한다.
또 다른 양상에 있어서, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 상기 트랜스멤브레인 α-헬릭스 2 은 SEQ ID NO : 2 를 포함하며, 상기 폴리펩티드는 셀로덱스트린 운반체인 숙주 세포를 준비하고, 숙주 세포가 재조합 폴리뉴클레오티드를 포함하지 않는 세포 보다, 배지에서 더 빠른 비율로 성장하는, 셀로덱스트린을 함유하는 배지에서 숙주 세포를 배양하는 것을 포함하는, 세포의 성장을 증가시키는 방법을 포함한다.
또 다른 양상에 있어서, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 상기 트랜스멤브레인 α-헬릭스 2 및 상기 트랜스멤브레인 α-헬릭스 3 을 연결하는 루프(loop)은 SEQ ID NO : 3 를 포함하며, 상기 폴리펩티드는 셀로덱스트린 운반체인 숙주 세포를 준비하고, 숙주 세포가 재조합 폴리뉴클레오티드를 포함하지 않는 세포 보다, 배지에서 더 빠른 비율로 성장하는, 셀로덱스트린을 함유하는 배지에서 숙주 세포를 배양하는 것을 포함하는, 세포의 성장을 증가시키는 방법을 포함한다.
또 다른 양상에 있어서, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 상기 트랜스멤브레인 α-헬릭스 5 은 SEQ ID NO : 4 를 포함하며, 상기 폴리펩티드는 셀로덱스트린 운반체인 숙주 세포를 준비하고, 숙주 세포가 재조합 폴리뉴클레오티드를 포함하지 않는 세포 보다, 배지에서 더 빠른 비율로 성장하는, 셀로덱스트린을 함유하는 배지에서 숙주 세포를 배양하는 것을 포함하는, 세포의 성장을 증가시키는 방법을 포함한다.
또 다른 양상에 있어서, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 상기 트랜스멤브레인 α-헬릭스 6 은 SEQ ID NO : 5 를 포함하며, 상기 폴리펩티드는 셀로덱스트린 운반체인 숙주 세포를 준비하고, 숙주 세포가 재조합 폴리뉴클레오티드를 포함하지 않는 세포 보다, 배지에서 더 빠른 비율로 성장하는, 셀로덱스트린을 함유하는 배지에서 숙주 세포를 배양하는 것을 포함하는, 세포의 성장을 증가시키는 방법을 포함한다.
또 다른 양상에 있어서, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 상기 트랜스멤브레인 α-헬릭스 6 및 상기 트랜스멤브레인 α-헬릭스 7 사이의 서열은 SEQ ID NO : 6 를 포함하며, 상기 폴리펩티드는 셀로덱스트린 운반체인 숙주 세포를 준비하고, 숙주 세포가 재조합 폴리뉴클레오티드를 포함하지 않는 세포 보다, 배지에서 더 빠른 비율로 성장하는, 셀로덱스트린을 함유하는 배지에서 숙주 세포를 배양하는 것을 포함하는, 세포의 성장을 증가시키는 방법을 포함한다.
또 다른 양상에 있어서, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 상기 트랜스멤브레인 α-헬릭스 7 은 SEQ ID NO : 7 를 포함하며, 상기 폴리펩티드는 셀로덱스트린 운반체인 숙주 세포를 준비하고, 숙주 세포가 재조합 폴리뉴클레오티드를 포함하지 않는 세포 보다, 배지에서 더 빠른 비율로 성장하는, 셀로덱스트린을 함유하는 배지에서 숙주 세포를 배양하는 것을 포함하는, 세포의 성장을 증가시키는 방법을 포함한다.
또 다른 양상에 있어서, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 상기 트랜스멤브레인 α-헬릭스 10, 상기 트랜스멤브레인 α-헬릭스 11 및 이들 사이의 서열은 SEQ ID NO : 8 을 포함하며, 상기 폴리펩티드는 셀로덱스트린 운반체인 숙주 세포를 준비하고, 숙주 세포가 재조합 폴리뉴클레오티드를 포함하지 않는 세포 보다, 배지에서 더 빠른 비율로 성장하는, 셀로덱스트린을 함유하는 배지에서 숙주 세포를 배양하는 것을 포함하는, 세포의 성장을 증가시키는 방법을 포함한다.
세포의 성장을 증가시키는 전술한 양상 중 어떠한 것과 결합할 수 있는 특정 실시형태에 있어서, 상기 폴리펩티드는 NCU00801 또는 NCU08114 와 적어도 29 %, 적어도 30 %, 적어도 35 %, 적어도 40 %, 적어도 45 %, 적어도 50 %, 적어도 55 %, 적어도 60 %, 적어도 65 %, 적어도 70 %, 적어도 75 %, 적어도 80 %, 적어도 85 %, 적어도 90 %, 적어도 95 %, 적어도 99 % 또는 적어도 100 % 의 아미노산 동일성을 갖는다. 전술한 실시형태 중 어떠한 것과 결합할 수 있는 특정 실시형태에 있어서, 숙주 세포는 내인성 또는 두 번째 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리뉴클레오티드는 β-글루코시다아제(β-glucosidase)의 적어도 촉매 도메인(catalytic domain)을 코드한다. β-글루코시다아제의 적어도 촉매 도메인을 코드하는, 내인성 또는 두 번째 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 갖는 전술한 실시형태와 결합할 수 있는 특정 실시형태에 있어서, β-글루코시다아제는 네우로스포라 크라사(Neurospora crassa)로부터 온 것이다. 네우로스포라 크라사로부터의 β-글루코시다아제의 적어도 하나의 촉매 도메인을 코드하는, 내인성 또는 두 번째 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 갖는 전술한 실시형태와 결합할 수 있는 특정 실시형태에 있어서, β-글루코시다아제는 NCU00130 에 의해 코드되었다.
또 다른 양상에 있어서, 본 발명은, 셀로덱스트린 운반체를 코딩하는(encoding) 첫 번째 재조합 폴리뉴클레오티드(first recombinant polynucleotide) 및 β-글루코시다아제의 촉매 도메인(catalytic domain)을 코딩하는 두 번째 재조합 폴리뉴클레오티드(second recombinant polynucleotide)를 포함하는 숙주 세포를 제공하고, 재조합 폴리뉴클레오티드의 발현이 셀룰로스-유도 당(cellulose-derived sugar) 및 헤미셀룰로스-유도 당(hemicellulose-derived sugar)의 공동-발효(co-fermentation)를 가능하게 하는, 셀룰로스-유도 당 및 헤미셀룰로스-유도 당을 포함하는 배지에 상기 숙주 세포를 배양시킴을 포함하는, 셀룰로스-유도 및 헤미셀룰로스-유도 당을 공동-발효시키는 방법을 포함한다. 특정 실시형태에 있어서, 첫 번째 재조합 폴리뉴클레오티드는 트랜스 멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하고, 트랜스멤브레인 α-헬릭스 1 은 SEQ ID NO: 1 을 포함한다. 특정 실시형태에 있어서, 첫 번째 재조합 폴리뉴클레오티드는 트랜스 멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하고, 트랜스멤브레인 α-헬릭스 2 은 SEQ ID NO: 2 를 포함한다. 특정 실시형태에 있어서, 첫 번째 재조합 폴리뉴클레오티드는 트랜스 멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하고, 트랜스멤브레인 α-헬릭스 2 및 트랜스멤브레인 α-헬릭스 3 을 연결하는 루프는 SEQ ID NO: 3 을 포함한다. 특정 실시형태에 있어서, 첫 번째 재조합 폴리뉴클레오티드는 트랜스 멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하고, 트랜스멤브레인 α-헬릭스 5 는 SEQ ID NO: 4 를 포함한다. 특정 실시형태에 있어서, 첫 번째 재조합 폴리뉴클레오티드는 트랜스 멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하고, 트랜스멤브레인 α-헬릭스 6 는 SEQ ID NO: 5 를 포함한다. 특정 실시형태에 있어서, 첫 번째 재조합 폴리뉴클레오티드는 트랜스 멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하고, 트랜스멤브레인 α-헬릭스 6 및 트랜스멤브레인 α-헬릭스 7 사이의 서열은 SEQ ID NO: 6 을 포함한다. 특정 실시형태에 있어서, 첫 번째 재조합 폴리뉴클레오티드는 트랜스 멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하고, 트랜스멤브레인 α-헬릭스 7 은 SEQ ID NO: 7 을 포함한다. 특정 실시형태에 있어서, 첫 번째 재조합 폴리뉴클레오티드는 트랜스 멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하고, 트랜스멤브레인 α-헬릭스 10, 트랜스멤브레인 α-헬릭스 11 및 이들 사이의 서열은 SEQ ID NO: 8 을 포함한다. 전술한 실시형태 중 어떠한 것과 결합할 수 있는 특정 실시형태에 있어서, 상기 폴리펩티드는 NCU00801 또는 NCU08114 와 적어도 29 %, 적어도 30 %, 적어도 35 %, 적어도 40 %, 적어도 45 %, 적어도 50 %, 적어도 55 %, 적어도 60 %, 적어도 65 %, 적어도 70 %, 적어도 75 %, 적어도 80 %, 적어도 85 %, 적어도 90 %, 적어도 95 %, 적어도 99 % 또는 적어도 100 % 의 아미노산 동일성을 갖는다. 전술한 실시형태 중 어떠한 것과 결합할 수 있는 특정 실시형태에 있어서, β-글루코시다아제는 네우로스포라 크라사(Neurospora crassa)로부터 온 것이다. 네우로스포라 크라사로부터의 β-글루코시다아제의 촉매 도메인을 코딩하는 두 번째 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 갖는 전술한 실시형태와 결합할 수 있는 특정 실시형태에서, β-글루코시다아제는 NCU00130 에 의해 코드되었다. 전술한 실시형태 중 어떠한 것과 결합할 수 있는 특정 실시형태에 있어서, 숙주 세포는 하나 또는 그 이상의 재조합 폴리뉴클레오티드를 추가로 포함하고, 상기 하나 또는 그 이상의 폴리뉴클레오티드는 오탄당 이용(pentose utilization)에 포함되는 하나 또는 그 이상의 효소를 코드한다. 하나 또는 그 이상의 재조합 폴리뉴클레오티드를 추가로 포함하는 숙주 세포를 갖되, 상기 하나 또는 그 이상의 재조합 폴리뉴클레오티드는 오탄당 이용에 포함되는 하나 또는 그 이상의 효소를 코드하는, 전술한 실시형태와 결합할 수 있는 특정 실시형태에서, 하나 또는 그 이상의 효소는, L-아라비노스 이성질화효소(L-arabinose isomerase), L-리블로인산화효소(L- ribulokinase), L-리블로스-5-P 4 에피머화효소(L- ribulose-5-P 4 epimerase), 자일로스 이성질화효소(xylose isomerase), 자일루로키나아제(xylulokinase), 알도스 환원효소(aldose reductase), L-아라비니톨 4-탈수소효소(L-arabinitol 4- dehydrogenase), L-자일루로스 환원효소(L- xylulose reductase) 및 자일리톨 탈수소효소로 이루어진 하나 또는 그 이상의 군으로부터 선택한 것이다. 전술한 실시형태 중 어떠한 것과 결합할 수 있는 특정 실시형태에 있어서, 숙주 세포는 세 번째 재조합 폴리뉴클레오티드를 추가로 포함하되, 상기 세 번째 재조합 폴리뉴클레오티드는 오탄당 운반체를 코드한다. 숙주 세포가 추가적으로 세 번째 재조합 폴리뉴클레오티드를 포함하되, 상기 세 번째 재조합 폴리뉴클레오티드는 오탄당 운반체를 코드하는 전술한 실시형태와 결합할 수 있는 특정 실시형태에서, 오탄당 운반체는 NCU00821, NCU04963, NCU06138, STL12/XUT6, SUT2, SUT3, XUT1 및 XUT3 으로 이루어진 군으로부터 선택한 것이다. 전술한 실시형태 중 어떠한 것과 결합할 수 있는 특정 실시형태에서, 상기 셀룰로스-유도 당은 셀로비오스, 셀로트리오스 및 셀로테트라오스로 이루어진 군으로부터 선택된 것이고, 헤미셀룰로스-유도 당은 자일로스이다.
또 다른 양상에 있어서, 본 발명은, 숙주 세포가 트랜스 멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하고, 상기 트랜스멤브레인 α-헬릭스 1 은 SEQ ID NO: 1 을 포함하고, 폴리펩티드는 탄화수소 또는 탄화수소 유도체를 합성하기 위해 숙주 세포 내로 셀로덱스트린을 수송하는, 숙주 세포를 제공하는 것을 포함하고, 재조합 폴리뉴클레오티드의 발현으로 상기 세포 내로 셀로덱스트린의 수송이 증가되는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 셀로덱스트린 또는 셀로덱스트린의 근원(source)을 포함하는 배지에서 숙주 세포를 배양하는 것을 포함하는, 숙주 세포에 의해 탄화수소(hydrocarbons) 또는 탄화수소 유도체(hydrocarbon derivatives)의 합성을 증가시키는 방법을 포함한다.
또 다른 양상에 있어서, 본 발명은, 숙주 세포가 트랜스 멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하고, 상기 트랜스멤브레인 α-헬릭스 2 는 SEQ ID NO: 2 를 포함하고, 폴리펩티드는 탄화수소 또는 탄화수소 유도체를 합성하기 위해 숙주 세포 내로 셀로덱스트린을 수송하는, 숙주 세포를 제공하는 것을 포함하고, 재조합 폴리뉴클레오티드의 발현으로 상기 세포 내로 셀로덱스트린의 수송이 증가되는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 셀로덱스트린 또는 셀로덱스트린의 근원(source)을 포함하는 배지에서 숙주 세포를 배양하는 것을 포함하는, 숙주 세포에 의해 탄화수소(hydrocarbons) 또는 탄화수소 유도체(hydrocarbon derivatives)의 합성을 증가시키는 방법을 포함한다.
또 다른 양상에 있어서, 본 발명은, 숙주 세포가 트랜스 멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하고, 상기 트랜스멤브레인 α-헬릭스 2 및 상기 트랜스멤브레인 α-헬릭스 3 을 연결하는 루프는 SEQ ID NO: 3 을 포함하고, 폴리펩티드는 탄화수소 또는 탄화수소 유도체를 합성하기 위해 숙주 세포 내로 셀로덱스트린을 수송하는, 숙주 세포를 제공하는 것을 포함하고, 재조합 폴리뉴클레오티드의 발현으로 상기 세포 내로 셀로덱스트린의 수송이 증가되는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 셀로덱스트린 또는 셀로덱스트린의 근원(source)을 포함하는 배지에서 숙주 세포를 배양하는 것을 포함하는, 숙주 세포에 의해 탄화수소(hydrocarbons) 또는 탄화수소 유도체(hydrocarbon derivatives)의 합성을 증가시키는 방법을 포함한다.
또 다른 양상에 있어서, 본 발명은, 숙주 세포가 트랜스 멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하고, 상기 트랜스멤브레인 α-헬릭스 5 는 SEQ ID NO: 4 를 포함하고, 폴리펩티드는 탄화수소 또는 탄화수소 유도체를 합성하기 위해 숙주 세포 내로 셀로덱스트린을 수송하는, 숙주 세포를 제공하는 것을 포함하고, 재조합 폴리뉴클레오티드의 발현으로 상기 세포 내로 셀로덱스트린의 수송이 증가되는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 셀로덱스트린 또는 셀로덱스트린의 근원(source)을 포함하는 배지에서 숙주 세포를 배양하는 것을 포함하는, 숙주 세포에 의해 탄화수소(hydrocarbons) 또는 탄화수소 유도체(hydrocarbon derivatives)의 합성을 증가시키는 방법을 포함한다.
또 다른 양상에 있어서, 본 발명은, 숙주 세포가 트랜스 멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하고, 상기 트랜스멤브레인 α-헬릭스 6 은 SEQ ID NO: 5 를 포함하고, 폴리펩티드는 탄화수소 또는 탄화수소 유도체를 합성하기 위해 숙주 세포 내로 셀로덱스트린을 수송하는, 숙주 세포를 제공하는 것을 포함하고, 재조합 폴리뉴클레오티드의 발현으로 상기 세포 내로 셀로덱스트린의 수송이 증가되는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 셀로덱스트린 또는 셀로덱스트린의 근원(source)을 포함하는 배지에서 숙주 세포를 배양하는 것을 포함하는, 숙주 세포에 의해 탄화수소(hydrocarbons) 또는 탄화수소 유도체(hydrocarbon derivatives)의 합성을 증가시키는 방법을 포함한다.
또 다른 양상에 있어서, 본 발명은, 숙주 세포가 트랜스 멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하고, 상기 트랜스멤브레인 α-헬릭스 6 및 상기 트랜스멤브레인 α-헬릭스 7 사이의 서열은 SEQ ID NO: 6 을 포함하고, 폴리펩티드는 탄화수소 또는 탄화수소 유도체를 합성하기 위해 숙주 세포 내로 셀로덱스트린을 수송하는, 숙주 세포를 제공하는 것을 포함하고, 재조합 폴리뉴클레오티드의 발현으로 상기 세포 내로 셀로덱스트린의 수송이 증가되는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 셀로덱스트린 또는 셀로덱스트린의 근원(source)을 포함하는 배지에서 숙주 세포를 배양하는 것을 포함하는, 숙주 세포에 의해 탄화수소(hydrocarbons) 또는 탄화수소 유도체(hydrocarbon derivatives)의 합성을 증가시키는 방법을 포함한다.
또 다른 양상에 있어서, 본 발명은, 숙주 세포가 트랜스 멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하고, 상기 트랜스멤브레인 α-헬릭스 7 은 SEQ ID NO: 7 을 포함하고, 폴리펩티드는 탄화수소 또는 탄화수소 유도체를 합성하기 위해 숙주 세포 내로 셀로덱스트린을 수송하는, 숙주 세포를 제공하는 것을 포함하고, 재조합 폴리뉴클레오티드의 발현으로 상기 세포 내로 셀로덱스트린의 수송이 증가되는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 셀로덱스트린 또는 셀로덱스트린의 근원(source)을 포함하는 배지에서 숙주 세포를 배양하는 것을 포함하는, 숙주 세포에 의해 탄화수소(hydrocarbons) 또는 탄화수소 유도체(hydrocarbon derivatives)의 합성을 증가시키는 방법을 포함한다.
또 다른 양상에 있어서, 본 발명은, 숙주 세포가 트랜스 멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하고, 상기 트랜스멤브레인 α-헬릭스 10, 상기 트랜스멤브레인 α-헬릭스 11 및 이들 사이의 서열은 SEQ ID NO: 8 을 포함하고, 폴리펩티드는 탄화수소 또는 탄화수소 유도체를 합성하기 위해 숙주 세포 내로 셀로덱스트린을 수송하는, 숙주 세포를 제공하는 것을 포함하고, 재조합 폴리뉴클레오티드의 발현으로 상기 세포 내로 셀로덱스트린의 수송이 증가되는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 셀로덱스트린 또는 셀로덱스트린의 근원(source)을 포함하는 배지에서 숙주 세포를 배양하는 것을 포함하는, 숙주 세포에 의해 탄화수소(hydrocarbons) 또는 탄화수소 유도체(hydrocarbon derivatives)의 합성을 증가시키는 방법을 포함한다.
탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 전술한 양상 중 어떠한 것과 결합할 수 있는 특정 실시형태에서, 상기 폴리펩티드는 NCU00801 또는 NCU08114 와 적어도 29 %, 적어도 30 %, 적어도 35 %, 적어도 40 %, 적어도 45 %, 적어도 50 %, 적어도 55 %, 적어도 60 %, 적어도 65 %, 적어도 70 %, 적어도 75 %, 적어도 80 %, 적어도 85 %, 적어도 90 %, 적어도 95 %, 적어도 99 % 또는 적어도 100 % 의 아미노산 동일성을 갖는다. 전술한 실시형태 중 어떠한 것과 결합할 수 있는 특정 실시형태에서, 숙주 세포는 추가로 두 번째 재조합 폴리뉴클레오티드를 포함하되, 상기 폴리뉴클레오티드는 β-글루코시다아제의 적어도 촉매 도메인을 코드한다. 숙주 세포가 추가로 두 번째 재조합 폴리뉴클레오티드를 포함하되, 상기 폴리뉴클레오티드가 β-글루코시다아제의 적어도 촉매 도메인을 코드하는 전술한 실시형태와 결합할 수 있는 특정 실시형태에서, β-글루코시다아제는 네우로스포라 크라사(Neurospora crassa)로부터 온 것이다. 숙주 세포가 추가로 두 번째 재조합 폴리뉴클레오티드를 포함하되, 상기 폴리뉴클레오티드가 네우로스포라 크라사로부터의 β-글루코시다아제의 적어도 촉매 도메인을 코드하는, 전술한 실시형태와 결합할 수 있는 특정 실시형태에서, β-글루코시다아제는 NCU00130 에 의해 코드되었다. 전술한 실시형태 중 어떠한 것과 결합할 수 있는 특정 실시형태에서, 셀로덱스트린 근원은 셀룰로스를 포함한다. 전술한 실시형태 중 어떠한 것과 결합할 수 있는 특정 실시형태에서, 탄화수소 또는 탄화수소 유도체는 연료(fuel)로서 사용될 수 있다. 탄화수소 또는 탄화수소 유도체는 연료로서 사용될 수 있는 전술한 실시형태와 결합할 수 있는 특정 실시형태에서, 탄화수소 또는 탄화수소 유도체는 에탄올을 포함한다. 탄화수소 또는 탄화수소 유도체는 연료로서 사용될 수 있는 전술한 실시형태와 결합할 수 있는 특정 실시형태에서, 탄화수소 또는 탄화수소 유도체는 부탄올(butanol)을 포함한다.
전술한 양상 중 어떠한 것과 결합할 수 있는 특정 실시형태에서, 상기 배지는 변경된 유기체(altered organism)로부터 셀룰라아제-함유 효소 혼합물(cellulase-containing enzyme mixture)을 포함하되, 상기 셀룰라아제-함유 혼합물(cellulase-containing mixture)은 변경되지 않는 유기체(unaltered organism)로부터의 셀룰라아제-함유 혼합물과 비교하여 감소된 β-글루코시다아제 활성도를 갖는다. 전술한 양상 중 어떠한 것과 결합할 수 있는 특정 실시형태에서, 상기 숙주 세포는, 사카로마이시스 ( Saccharomyces sp .), 사카로미세스 세레비시아( Saccharomyces cerevisiae ), 사카로미세스 모나센시스( Saccharomyces monacensis), 사카로미세스 바야누스 ( Saccharomyces bayanus ), 사카로미세스 파스토리아누스( Saccharomyces pastorianus ), 사카로미세스 칼스베르겐시스( Saccharomyces carlsbergensis ), 사카로미세스 폼베 ( Saccharomyces pombe ), 클루이베르마이세스( Kluyveromyces sp .), 클루이베르마이세스 마르시암우스( Kluyveromyces marxiamus ), 클루이베르마이세스 락티스( Kluyveromyces lactis), 클루이베르마이세스 프라길리스 ( Kluyveromyces fragilis ), 피치아 스티피 티스( Pichia stipitis ), 스포로트리쿰 서머파일 ( Sporotrichum thermophile ), 칸디 다 쉬하테( Candida shehatae ), 칸디다 트로피칼리스 ( Candida tropicalis ), 네우로 스포라 크라사 ( Neurospora crassa ), 자이모나스 모빌리스( Zymomonas mobilis ), 클로스트리듐( Clostridium sp .), 클로스트리듐 파이토퍼멘탄스( Clostridium phytofermentans), 클로스트리듐 써모셀럼 ( Clostridium thermocellum ), 클로스트리듐 베이저린키이( Clostridium beijerinckii ), 클로스트리듐 아세토부틸리쿰( Clostridium acetobutylicum ), 무렐라 서모아세티카 ( Moorella thermoacetica ), 대장균( Escherichia coli ), 크레브시엘라 옥시토카 ( Klebsielle oxytoca ), 서모안아에로박테리움 사카로락티쿰 ( Thermoanaerobacterium saccharolyticum ) 및 고초균( Bacillus subtilis )으로 이루어진 군으로부터 선택한 것이다. 전술한 양상 중 어떠한 것과 결합할 수 있는 특정 실시형태에서, 상기 셀로덱스트린은, 셀로비오스, 셀로트리오스 및 셀로테트라오스로 이루어진 하나 또는 그 이상의 군으로부터 선택된 것이다.
또 다른 양상은 트랜스 멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12, 세포 내의 N-말단(intracellular N-terminus), 세포 내의 C-말단, 및 트랜스 멤브레인 α-헬릭스 1 에서의 SEQ ID NO : 1, 트랜스 멤브레인 α-헬릭스 2 에서의 SEQ ID NO : 2, 트랜스 멤브레인 α-헬릭스 2 및 트랜스 멤브레인 α-헬릭스 3 을 연결하는 루프에서의 SEQ ID NO : 3, 트랜스 멤브레인 α-헬릭스 5 에서의 SEQ ID NO : 4, 트랜스 멤브레인 α-헬릭스 6 에서의 SEQ ID NO : 5, 트랜스 멤브레인 α-헬릭스 6 및 트랜스 멤브레인 α-헬릭스 7 사이의 서열에서 SEQ ID NO : 6, 트랜스 멤브레인 α-헬릭스 7 에서의 SEQ ID NO : 7 및 트랜스 멤브레인 α-헬릭스 10, 트랜스 멤브레인 α-헬릭스 11 및 이들 사이의 서열에서 SEQ ID NO : 8 로 이루어진 군으로부터 선택된 서열을 갖는 폴리펩티드를 코딩하는 재조합 폴레뉴클레오티드를 포함하는 숙주 세포를 포함하되, 상기 폴리펩티드는 셀로덱스트린 운반체이다. 특정 실시형태에 있어서, 상기 폴리펩티드는 NCU00801 또는 NCU08114 와 적어도 29 %, 적어도 30 %, 적어도 35 %, 적어도 40 %, 적어도 45 %, 적어도 50 %, 적어도 55 %, 적어도 60 %, 적어도 65 %, 적어도 70 %, 적어도 75 %, 적어도 80 %, 적어도 85 %, 적어도 90 %, 적어도 95 %, 적어도 99 % 또는 적어도 100 % 의 아미노산 동일성을 갖는다. 전술한 실시형태 중 어떤 것과 결합할 수 있는 특정 실시형태에서, 상기 숙주 세포는 추가로 두 번째 재조합 폴리뉴클레오티드를 포함하되, 상기 두 번째 재조합 폴리뉴클레오티드는 β-글루코시다아제의 촉매 도메인을 코드한다. 상기 두 번째 재조합 폴리뉴클레오티드는 β-글루코시다아제의 촉매 도메인을 코드하는, 두 번째 재조합 폴리뉴클레오티드를 추가로 포함하는 숙주 세포를 갖는 전술한 실시형태와 결합할 수 있는 특정 실시형태에서, β-글루코시다아제는 네우로스포라 크라사(Neurospora crassa)로부터 온 것이다. 네우로스포라 크라사로부터의 β-글루코시다아제의 촉매 도메인을 코드하는 두 번째 재조합 폴리뉴클레오티드를 추가로 포함하는 숙주세포를 갖는 전술한 실시형태와 결합할 수 있는 특정 실시형태에서, β-글루코시다아제는 NCU00130 에 의해 코드되었다. 전술한 실시형태 중 어떠한 것과 결합할 수 있는 특정 실시형태에서, 상기 숙주 세포는 추가로 하나 또는 그 이상의 재조합 폴리뉴클레오티드를 포함하되, 상기 하나 또는 그 이상의 폴리뉴클레오티드는 오탄당 이용에 포함되는 하나 또는 그 이상의 효소를 코드한다. 상기 하나 또는 그 이상의 폴리뉴클레오티드는 오탄당 이용에 포함되는 하나 또는 그 이상의 효소를 코드하고, 하나 또는 그 이상의 재조합 폴리뉴클레오티드를 추가로 포함하는 숙주 세포를 갖는 전술한 실시형태와 결합할 수 있는 특정 실시형태에서, 하나 또는 그 이상의 효소는, L-아라비노스 이성질화효소, L-리블로인산화효소, L-리블로스-5-P 4 에피머화효소, 자일로스 이성질화효소, 자일루로키나아제, 알도스 환원효소, L-아라비니톨 4-탈수소효소, L-자일루로스 환원효소 및 자일리톨 탈수소효소로 이루어진 하나 또는 그 이상의 군으로부터 선택된 것이다. 전술한 실시형태 중 어떠한 것과 결합할 수 있는 특정 실시형태에서, 상기 숙주 세포는 추가로 세 번째 재조합 폴리뉴클레오티드를 포함하되, 상기 세 번째 재조합 폴리뉴클레오티드는 오탄당 운반체를 코드한다. 오탄당 운반체를 코드하는 세 번째 재조합 폴리뉴클레오티드를 추가로 포함하는 숙주 세포를 갖는 전술한 실시형태와 결합할 수 있는 특정 실시형태에서, 오탄당 운반체는 NCU00821, NCU04963, NCU06138, STL12/XUT6, SUT2, SUT3, XUT1 및 XUT3 으로 이루어진 군으로부터 선택된 것이다.
전술한 양상 중 어떠한 것과 결합할 수 있는 특정 실시형태에서, 숙주 세포는 하나 또는 그 이상의 재조합 폴리뉴클레오티드에 작동가능하게 연결된(operably linked) 하나 또는 그 이상의 유도성 프로모터(inducible promoters)를 추가로 포함한다.
또 다른 양상은 NCU00821 및 STL12/XUT6 으로 이루어진 군으로부터 선택된 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 포함하되, 상기 폴리펩티드는 세포 내로 자일로스를 수송한다.
또 다른 양상은 XUT1 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 포함하되, 상기 폴리펩티드는 세포 내로 아라비노스를 수송한다.
또 다른 양상은 NCU06138 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 포함하되, 상기 폴리펩티드는 세포 내로 아라비노스 및 글루코스를 수송한다.
또 다른 양상은 SUT2, SUT3 및 XUT3 로 이루어진 군으로부터 선택된 폴리뉴클레오티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 포함하되, 상기 폴리펩티드는 세포 내로 자일로스 및 글루코스를 수송한다.
또 다른 양상은 NCU04963 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 포함하되, 상기 폴리펩티드는 세포 내로 자일로스, 아라비노스 및 글루코스를 수송한다.
오탄당 운반체를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 갖는 전술한 양상 중 어떠한 것과 결합할 수 있는 특정 실시형태에서, 상기 숙주 세포는 하나 또는 그 이상의 재조합 폴리뉴클레오티드를 추가로 포함하되, 상기 하나 또는 그 이상의 폴리뉴클레오티드는 오탄당 이용에 포함되는 하나 또는 그 이상의 효소를 코드한다. 오탄당 이용에 포함되는 하나 또는 그 이상의 효소를 코드하는 하나 또는 그 이상의 폴리뉴클레오티드를 추가로 포함하는 숙주 세포를 갖는 전술한 실시형태와 결합될 수 있는 특정 실시형태에서, 하나 또는 그 이상의 효소는, L-아라비노스 이성질화효소, L-리블로인산화효소, L-리블로스-5-P 4 에피머화효소, 자일로스 이성질화효소, 자일루로키나아제, 알도스 환원효소, L-아라비니톨 4-탈수소효소, L-자일루로스 환원효소 및 자일리톨 탈수소효소로 이루어진 하나 또는 그 이상의 군으로부터 선택된 것이다.
또 다른 양상에서 본 발명은, NCU00821 및 STL12/XUT6 로 이루어진 군으로부터 선택된 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하는 것을 포함하고, 재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로 자일로스 수송이 증가되는, 재조합 폴리뉴클레오티드가 발현될 정도까지 세포를 배양하는 것을 포함하는, 세포 내로 자일로스의 수송을 증가시키는 방법을 포함한다.
또 다른 양상에서 본 발명은, XUT1 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하는 것을 포함하고, 재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로 아라비노스 수송이 증가되는, 재조합 폴리뉴클레오티드가 발현될 정도까지 세포를 배양하는 것을 포함하는, 세포 내로 아라비노스의 수송을 증가시키는 방법을 포함한다.
또 다른 양상에서 본 발명은, NCU06138 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하는 것을 포함하고, 재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로 아라비노스 또는 글루코스 수송이 증가되는, 재조합 폴리뉴클레오티드가 발현될 정도까지 세포를 배양하는 것을 포함하는, 세포 내로 아라비노스 또는 글루코스의 수송을 증가시키는 방법을 포함한다.
또 다른 양상에서 본 발명은, SUT2, SUT3 및 XUT3 으로 이루어진 군으로부터 선택된 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하는 것을 포함하고, 재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로 자일로스 또는 글루코스 수송이 증가되는, 재조합 폴리뉴클레오티드가 발현될 정도까지 세포를 배양하는 것을 포함하는, 세포 내로 자일로스 또는 글루코스의 수송을 증가시키는 방법을 포함한다.
또 다른 양상에서 본 발명은, NCU04963 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하는 것을 포함하고, 재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로 자일로스, 아라비노스 또는 글루코스 수송이 증가되는, 재조합 폴리뉴클레오티드가 발현될 정도까지 세포를 배양하는 것을 포함하는, 세포 내로 자일로스, 아라비노스 또는 글루코스의 수송을 증가시키는 방법을 포함한다.
세포 내로 자일로스, 아라비노스 또는 글루코스의 수송을 증가시키는 전술한 양상 중 어떠한 것과 결합할 수 있는 특정 실시형태에서, 방법은 추가적으로 하나 또는 그 이상의 재조합 폴리뉴클레오티드를 포함하되, 상기 하나 또는 그 이상의 폴리뉴클레오티드는 오탄당 이용에 포함되는 하나 또는 그 이상의 효소를 코드한다. 하나 또는 그 이상의 폴리뉴클레오티드가 오탄당 이용에 포함되는 하나 또는 그 이상의 효소를 코드하는, 추가로 하나 또는 그 이상의 재조합 폴리뉴클레오티드를 포함하는 방법을 갖는 전술한 실시형태와 결합될 수 있는 특정 실시형태에서, 하나 또는 그 이상의 효소는, L-아라비노스 이성질화효소, L-리블로인산화효소, L-리블로스-5-P 4 에피머화효소, 자일로스 이성질화효소, 자일루로키나아제, 알도스 환원효소, L-아라비니톨 4-탈수소효소, L-자일루로스 환원효소 및 자일리톨 탈수소효소로 이루어진 하나 또는 그 이상 군으로부터 선택된 것이다.
또 다른 양상은, 숙주 세포는 NCU00821 및 STL12/XUT6 으로 이루어진 군으로부터 선택된 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리펩티드는 자일로스를 수송하는, 상기 숙주 세포를 제공하는 것을 포함하며, 상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 더 빠른 비율로 성장하는, 자일로스를 포함하는 배지에서 숙주 세포를 배양하는 것을 포함하는, 세포의 성장을 증가키는 방법을 포함한다.
또 다른 양상은, 숙주 세포는 XUT1 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리펩티드는 아라비노스를 수송하는, 상기 숙주 세포를 제공하는 것을 포함하며, 상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 더 빠른 비율로 성장하는, 아라비노스를 포함하는 배지에서 숙주 세포를 배양하는 것을 포함하는, 세포의 성장을 증가키는 방법을 포함한다.
또 다른 양상은, 숙주 세포는 NCU06138 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리펩티드는 아라비노스 및 글루코스를 수송하는, 상기 숙주 세포를 제공하는 것을 포함하며, 상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 더 빠른 비율로 성장하는, 아라비노스 또는 글루코스를 포함하는 배지에서 숙주 세포를 배양하는 것을 포함하는, 세포의 성장을 증가키는 방법을 포함한다.
또 다른 양상은, 숙주 세포는 SUT2, SUT3 및 XUT3 으로 이루어진 군으로부터 선택한 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리펩티드는 자일로스 및 글루코스를 수송하는, 상기 숙주 세포를 제공하는 것을 포함하며, 상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 더 빠른 비율로 성장하는, 자일로스 또는 글루코스를 포함하는 배지에서 숙주 세포를 배양하는 것을 포함하는, 세포의 성장을 증가키는 방법을 포함한다.
또 다른 양상은, 숙주 세포는 NCU04963 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리펩티드는 자일로스, 아라비노스 및 글루코스를 수송하는, 상기 숙주 세포를 제공하는 것을 포함하며, 상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 더 빠른 비율로 성장하는, 자일로스, 아라비노스 또는 글루코스를 포함하는 배지에서 숙주 세포를 배양하는 것을 포함하는, 세포의 성장을 증가키는 방법을 포함한다.
자일로스 및/또는 아라비노스 및/또는 글루코스를 수송하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 배양하여 세포의 성장을 증가시키는 전술한 양상과 결합될 수 있는 특정 실시형태에서, 숙주 세포는, 오탄당 이용에 포함되는 하나 또는 그 이상의 효소를 코딩하는 하나 또는 그 이상의 내인성 또는 재조합 폴리뉴클레오티드를 추가로 포함한다. 오탄당 이용에 포함되는 하나 또는 그 이상의 효소를 코딩하는 하나 또는 그 이상의 내인성 또는 재조합 폴리뉴클레오티드를 추가로 포함하는 숙주 세포를 갖는 전술한 실시형태와 결합될 수 있는 특정 실시형태에서, 하나 또는 그 이상의 효소는, L-아라비노스 이성질화효소, L-리블로인산화효소, L-리블로스-5-P 4 에피머화효소, 자일로스 이성질화효소, 자일루로키나아제, 알도스 환원효소, L-아라비니톨 4-탈수소효소, L-자일루로스 환원효소 및 자일리톨 탈수소효소로 이루어진 하나 또는 그 이상 군으로부터 선택된 것이다.
또 다른 양상은, NCU00821 및 STL12/XUT6 로 이루어진 군으로부터 선택된 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하되, 상기 폴리펩티드는 탄화수소 또는 탄화수소 유도체의 합성을 위해 숙주 세포 내로 자일로스를 수송하며, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 자일로스 또는 자일로스 근원을 포함하는 배지에서 숙주 세포를 배양하되, 상기 세포 내로의 자일로스의 수송은 재조합 폴리뉴클레오티드의 발현으로 증가되는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법을 포함한다.
또 다른 양상은, XUT1 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하되, 상기 폴리펩티드는 탄화수소 또는 탄화수소 유도체의 합성을 위해 숙주 세포 내로 아라비노스를 수송하며, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 아라비노스 또는 아라비노스 근원을 포함하는 배지에서 숙주 세포를 배양하되, 상기 세포 내로의 아라비노스의 수송은 재조합 폴리뉴클레오티드의 발현으로 증가되는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법을 포함한다.
또 다른 양상은, NCU06138 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하되, 상기 폴리펩티드는 탄화수소 또는 탄화수소 유도체의 합성을 위해 숙주 세포 내로 아라비노스 또는 글루코스를 수송하며, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 아라비노스 또는 글루코스 또는 아라비노스 또는 글루코스의 근원을 포함하는 배지에서 숙주 세포를 배양하되, 상기 세포 내로의 아라비노스 또는 글루코스의 수송은 재조합 폴리뉴클레오티드의 발현으로 증가되는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법을 포함한다.
또 다른 양상은, SUT2, SUT3 및 XUT3 으로 이루어진 군으로부터 선택된 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하되, 상기 폴리펩티드는 탄화수소 또는 탄화수소 유도체의 합성을 위해 숙주 세포 내로 자일로스 또는 글루코스를 수송하며, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 자일로스 또는 글루코스 또는 자일로스 또는 글루코스의 근원을 포함하는 배지에서 숙주 세포를 배양하되, 상기 세포 내로의 자일로스 또는 글루코스의 수송은 재조합 폴리뉴클레오티드의 발현으로 증가되는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법을 포함한다.
또 다른 양상은, NCU04963 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하되, 상기 폴리펩티드는 탄화수소 또는 탄화수소 유도체의 합성을 위해 숙주 세포 내로 자일로스, 아라비노스 또는 글루코스를 수송하며, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 자일로스, 아라비노스 또는 글루코스 또는 자일로스, 아라비노스 또는 글루코스의 근원을 포함하는 배지에서 숙주 세포를 배양하되, 상기 세포 내로의 자일로스, 아라비노스 또는 글루코스의 수송은 재조합 폴리뉴클레오티드의 발현으로 증가되는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법을 포함한다.
글루코스를 수송하는 펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 배양하여 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 전술한 양상 중 어떠한 것과 결합할 수 있는 특정 실시형태에서, 글루코스의 근원은 셀룰로스를 포함한다. 전술한 실시형태 중 어떠한 것과 결합할 수 있는 특정 실시형태에서, 자일로스 또는 아라비노스의 근원은 헤미셀룰로스를 포함한다. 전술한 실시형태 중 어떠한 것과 결합할 수 있는 특정 실시형태에서, 탄화수소 또는 탄화수소 유도체는 연료로서 사용될 수 있다. 탄화수소 또는 탄화수소 유도체는 연료로서 사용될 수 있는 것을 갖는 전술한 실시형태와 결합할 수 있는 특정 실시형태에서, 탄화수소 또는 탄화수소 유도체는 에탄올을 포함한다. 탄화수소 또는 탄화수소 유도체는 연료로서 사용될 수 있는 것을 갖는 전술한 실시형태와 결합할 수 있는 특정 실시형태에서, 탄화수소 또는 탄화수소 유도체는 부탄올을 포함한다.
전술한 실시형태 중 어떠한 것과 결합할 수 있는 특정 실시형태에서, 상기 숙주 세포는, 사카로마이시스 ( Saccharomyces sp .), 사카로미세스 세레비시아( Saccharomyces cerevisiae), 사카로미세스 모나센시스 ( Saccharomyces monacensis ), 사카로미세스 바야누스( Saccharomyces bayanus ), 사카로미세스 파스 토리아누스( Saccharomyces pastorianus ), 사카로미세스 칼스베르겐시스( Saccharomyces carlsbergensis ), 사카로미세스 폼베 ( Saccharomyces pombe ), 클루이베르마이세스( Kluyveromyces sp .), 클루이베르마이세스 마르시암우 스( Kluyveromyces marxiamus ), 클루이베르마이세스 락티스( Kluyveromyces lactis), 클루이베르마이세스 프라길리스 ( Kluyveromyces fragilis ), 피치아 스티피 티스( Pichia stipitis), 스포로트리쿰 서머파일 ( Sporotrichum thermophile ), 칸디다 쉬하테( Candida shehatae), 칸디다 트로피칼리스 ( Candida tropicalis ), 네우로스포라 크라사( Neurospora crassa), 자이모나스 모빌리스( Zymomonas mobilis ), 클로스트리듐( Clostridium sp .), 클로스트리듐 파이토퍼멘탄스( Clostridium phytofermentans ), 클로스트리듐 써모셀럼( Clostridium thermocellum), 클로스트리듐 베이저린키이 ( Clostridium beijerinckii ), 클로스트리듐 아세토부틸리쿰( Clostridium acetobutylicum ), 무렐라 서모아세티카 ( Moorella thermoacetica), 대장균( Escherichia coli ), 크레브시엘라 옥시토카( Klebsielle oxytoca), 서모안아에로박테리움 사카로락티쿰( Thermoanaerobacterium saccharolyticum ) 및 고초균( Bacillus subtilis )으로 이루어진 군으로부터 선택한 것이다.
또 다른 양상은, NCU07705 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하는 것을 포함하고, 숙주 세포가 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 더 빠른 비율로 성장하고, 셀로비오스를 포함하는 배지에서 세포를 배양하는 것을 포함하는, 세포의 성장을 증가시키는 방법을 포함한다. 특정 실시형태에서, 상기 숙주 세포는, 사카로마이시스( Saccharomyces sp .), 사카로미세스 세레비시아 ( Saccharomyces cerevisiae ), 사 카로미세스 모나센시스 ( Saccharomyces monacensis ), 사카로미세스 바야누스( Saccharomyces bayanus ), 사카로미세스 파스토리아누스( Saccharomyces pastorianus), 사카로미세스 칼스베르겐시스 ( Saccharomyces carlsbergensis ), 사카로미세스 폼베 ( Saccharomyces pombe ), 클루이베르마이세스 ( Kluyveromyces sp .), 클루이베르마이세스 마르시암우스 ( Kluyveromyces marxiamus ), 클루이베르마이세스 락티스( Kluyveromyces lactis ), 클루이베르마이세스 프라길리스( Kluyveromyces fragilis), 피치아 스티피티스 ( Pichia stipitis ), 스포로트리쿰 서머파일( Sporotrichum thermophile ), 칸디다 쉬하테 ( Candida shehatae ), 칸디다 트로피칼리스( Candida tropicalis ), 네우로스포라 크라사 ( Neurospora crassa ), 자이모나스 모빌리스( Zymomonas mobilis ), 클로스트리듐( Clostridium sp .), 클로스트리듐 파이토퍼멘탄스( Clostridium phytofermentans ), 클로스트리듐 써모셀럼( Clostridium thermocellum ), 클로스트리듐 베이저린키이( Clostridium beijerinckii), 클로스트리듐 아세토부틸리쿰 ( Clostridium acetobutylicum ), 무렐라 서모아세티카 ( Moorella thermoacetica ), 대장균( Escherichia coli ), 크레브시엘라 옥시토카 ( Klebsielle oxytoca ), 서모안아에로박테리움 사카로락티쿰( Thermoanaerobacterium saccharolyticum ) 및 고초균( Bacillus subtilis )으로 이루어진 군으로부터 선택한 것이다. 특정 실시형태에서, 숙주 세포는 재조합 폴리뉴클레오티드에 작동가능하게 연결된 유도성 프로모터를 추가로 포함한다. 특정 실시형태에서, 셀룰라아제의 발현은 재조합 폴리뉴클레오티드의 발현으로 숙주 세포에서 증가되었다.
또 다른 양상은, NCU05137 폴리펩티드를 코드하는 내인성 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하고, 내인성 폴리뉴클레오티드의 발현을 저해하며, 상기 숙주 세포는 상기 내인성 폴리뉴클레오티드의 발현을 저해하지 않는 세포보다, 배지에서 더 빠른 비율로 성장하는, 바이오매스 중합체를 포함하는 배지에서 세포를 배양하는 것을 포함하는, 바이오매스 중합체(biomass polymer)에서 세포의 성장을 증가시키는 방법을 포함한다. 특정 실시형태에서, 상기 숙주 세포는, 사카로마이시스( Saccharomyces sp .), 사카로미세스 세레비시아 ( Saccharomyces cerevisiae ), 사카로미세스 모나센시스 ( Saccharomyces monacensis ), 사카로미세스 바야누스 ( Saccharomyces bayanus ), 사카로미세스 파스토리아누스 ( Saccharomyces pastorianus ), 사카로미세스 칼스베르겐시스( Saccharomyces carlsbergensis ), 사카로미세스 폼베 ( Saccharomyces pombe ), 클루이베르마이세스( Kluyveromyces sp .), 클루이베르마이세스 마르시암우스( Kluyveromyces marxiamus), 클루이베르마이세스 락티스 ( Kluyveromyces lactis ), 클루이베르마이세스 프라길리스( Kluyveromyces fragilis ), 피치아 스티피티스 ( Pichia stipitis ), 스포로트리쿰 서머파일( Sporotrichum thermophile ), 칸디다 쉬하테 ( Candida shehatae ), 칸디다 트로피칼리스( Candida tropicalis ), 네우로스포라 크라사 ( Neurospora crassa ), 자이모나스 모빌리스( Zymomonas mobilis ), 클로스트리듐( Clostridium sp .), 클로스트리듐 파이토퍼멘탄스( Clostridium phytofermentans ), 클로스트리듐 써모셀럼( Clostridium thermocellum), 클로스트리듐 베이저린키이 ( Clostridium beijerinckii ), 클로스트리듐 아세토부틸리쿰( Clostridium acetobutylicum ), 무렐라 서모아세티카 ( Moorella thermoacetica ), 대장균( Escherichia coli ), 크레브시엘 라 옥시토카 ( Klebsielle oxytoca ), 서모안아에로박테리움 사카로락티쿰( Thermoanaerobacterium saccharolyticum ) 및 고초균( Bacillus subtilis )으로 이루어진 군으로부터 선택된 것이다. 특정 실시형태에서, 숙주 세포의 셀룰라아제 활성도는 내인성 폴리뉴클레오티드의 발현 저해로 증가되었다. 특정 실시형태에서, 숙주 세포의 헤미셀룰라아제 활성도는 내인성 폴리뉴클레오티드의 발현 저해로 증가되었다. 특정 실시형태에서, 내인성 폴리뉴클레오티드의 발현을 저해하는 것은 내인성 폴리뉴클레오티드를 포함하는 유전자의 돌연변이(mutating) 또는 결실(deleting)을 포함한다. 특정 실시형태에서, 바이오매스 중합체는 셀룰로스이다. 특정 실시형태에서, 바이오매스 중합체(biomass polymer)는 헤미셀룰로스이다.
또 다른 양상은, NCU01517, NCU09133 및 NCU10040 으로 이루어진 군으로부터 선택된 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하고, 숙주 세포가 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 더 빠른 비율로 성장하는, 헤미셀룰로스를 포함하는 배지에서 세포를 배양시키는 것을 포함하는, 세포의 성장을 증가시키는 방법을 포함한다. 특정 실시형태에서, 상기 숙주 세포는, 사카로마이시스( Saccharomyces sp .), 사카로미세스 세레비시아( Saccharomyces cerevisiae ), 사카로미세스 모나센시스( Saccharomyces monacensis), 사카로미세스 바야누스 ( Saccharomyces bayanus ), 사카로미세스 파스토리아누스( Saccharomyces pastorianus ), 사카로미세스 칼스베르겐시스( Saccharomyces carlsbergensis ), 사카로미세스 폼베 ( Saccharomyces pombe ), 클루이베르마이세스( Kluyveromyces sp .), 클루이베르마이세스 마르시암우스( Kluyveromyces marxiamus ), 클루이베르마이세스 락티스( Kluyveromyces lactis), 클루이베르마이세스 프라길리스 ( Kluyveromyces fragilis ), 피치아 스티피티스( Pichia stipitis ), 스포로트리쿰 서머파일 ( Sporotrichum thermophile ), 칸디다 쉬하테( Candida shehatae ), 칸디다 트로피칼리스 ( Candida tropicalis ), 네우로스포라 크라사 ( Neurospora crassa ), 자이모나스 모빌리스( Zymomonas mobilis ), 클로스트리듐( Clostridium sp .), 클로스트리듐 파이토퍼멘탄스( Clostridium phytofermentans), 클로스트리듐 써모셀럼 ( Clostridium thermocellum ), 클로스트리듐 베이저린키이( Clostridium beijerinckii ), 클로스트리듐 아세토부틸리쿰( Clostridium acetobutylicum ), 무렐라 서모아세티카 ( Moorella thermoacetica ), 대장균( Escherichia coli ), 크레브시엘라 옥시토카 ( Klebsielle oxytoca ), 서모안아에로박테리움 사카로락티쿰 ( Thermoanaerobacterium saccharolyticum ) 및 고초균( Bacillus subtilis )으로 이루어진 군으로부터 선택된 것이다. 특정 실시형태에서, 숙주 세포는 재조합 폴리뉴클레오티드에 작동가능하게 연결된 유도성 프로모터를 더 포함한다. 특정 실시형태에서, 숙주 세포의 헤미셀룰라아제 활성도는 재조합 폴리뉴클레오티드의 발현으로 증가되었다.
또 다른 양상은, 셀룰로스를 포함하는 조성물을 제공하고, 변경된 유기체(altered organism)로부터의 셀룰라아제-함유 효소 혼합물(cellulase-containing enzyme mixture)을 갖는 조성물(composition)과 접촉하되, 상기 셀룰라아제-함유 혼합물은, 변경되지 않는 유기체(unaltered organism)로부터의 셀룰라아제-함유 혼합물과 비교하여, 감소된 β-글루코시다아제 활성도를 갖고, 상기 셀룰로스는 셀룰라아제-함유 혼합물에 의해 분해되는, 셀룰로스 분해(degrading) 방법을 포함한다. 특정 실시형태에서, 상기 유기체는 β-글루코시다아제를 코딩하는 유전자의 돌연변이에 의해 변경되었다(altered). 특정 실시형태에서, 상기 유기체는 β-글루코시다아제의 발현의 감소에 의해 변경되었다. 전술한 실시형태 중 어떠한 것과 결합할 수 있는 특정 실시형태에서, 상기 유기체는 균류(fungus) 또는 세균(bacterium) 으로 이루어진 군으로부터 선택된 것이다. 균류(fungus) 또는 세균(bacterium) 으로 이루어진 군으로부터 선택된 상기 유기체를 갖는 전술한 실시형태 중 어떠한 것과 결합할 수 있는 특정 실시형태에서, 상기 유기체는 사상균(filamentous fungus)이다. 전술한 실시형태 중 어떠한 것과 결합할 수 있는 특정 실시형태에서, 셀룰로스는 식물 물질(plant material)로부터 온 것이다. 식물 물질(plant material)로부터 온 셀룰로스를 갖는 전술한 실시형태 중 어떠한 것과 결합할 수 있는 특정 실시형태에서, 식물 물질은 지팽이풀(switchgrass), 미스칸터스(Miscanthus), 왕겨(rice hull), 버개스(bagasse), 아마(flax), 대나무(bamboo), 사이잘(sisal), 마닐라삼섬유(abaca), 짚(straw), 잎(leaves), 잔디 부스러기(grass clippings), 옥수수대(corn stover), 옥수수 속대(corn cobs), 증류립(distillers grains), 콩과 식물(legume plants), 수수(sorghum), 사탕수수(sugar cane), 사탕무 펄프(sugar beet pulp), 우드칩(wood chips), 톱밥(sawdust) 및 바이오매스 크롭(biomass crops)으로 이루어진 군으로부터 선택된 것이다.
또 다른 양상은, NCU00801, NCU00988, NCU01231, NCU04963, NCU05519, NCU05853, NCU05897, NCU06138, NCU00809, NCU08114, NCU10021 및 표 15 에 나타낸 유전자 중의 어떠한 것으로 이루어진 군으로부터 선택된 서열에 의해 코드된 폴리펩티드를 코드하는 재조합 폴리뉴클레오티르를 포함하는 숙주 세포를 제공하고, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 화합물(compound)의 근원을 포함하는 배지에서 숙주 세포를 배양하는 것을 포함하되, 상기 화합물은 탄화수소 또는 탄화수소 유도체의 합성을 위한 기질(substrate)이고, 세포 내로 화합물의 수송은 재조합 폴리뉴클레오티드의 발현으로 증가된, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법을 포함한다. 특정 실시형태에서, 숙주 세포는, 사카로마이시스 ( Saccharomyces sp .), 대장균( Escherichia coli ), 자이모나스 모빌리스( Zymomonas mobilis ), 네우로스포라 크 라사 ( Neurospora crassa ), 칸디다 쉬하테( Candida shehatae ), 클로스트리듐( Clostridium sp .), 클로스트리듐 파이토퍼멘탄스( Clostridium phytofermentans ), 클로스트리듐 써모셀럼( Clostridium thermocellum), 무렐라 서모아세티카 ( Moorella thermoacetica ), 서모안아에로박테리움 사카로락티 쿰( Thermoanaerobacterium saccharolyticum ), 클레브시엘라 옥시토카(Klebsiella oxytoca) 및 피치아 스티피티스(Pichia stipitis)으로 이루어진 군으로부터 선택된 것이다. 특정 실시형태에서, 숙주 세포는 재조합 폴리뉴클레오티드에 작동가능하게 연결된 유도성 프로모터를 더 포함한다. 특정 실시형태에서, 재조합 폴리뉴클레오티드는, NCU00801, NCU00988, NCU01231, NCU04963, NCU05519, NCU05853, NCU05897, NCU06138, NCU00809, NCU08114, NCU10021, 및 표 15 에 나타낸 유전자의 어떠한 것으로 이루어진 군으로부터 선택된 서열을 코드하는 폴리펩티드와 적어도 50 % 의 아미노산 동일성을 갖는 폴리펩티드를 코드한다. 몇몇 실시형태에서, 탄화수소 또는 탄화수소 유도체는 연료로서 사용될 수 있다. 특정 실시형태에서, 배지는 셀룰로스를 포함한다. 다른 실시형태에서, 상기 배지는 헤미셀룰로스를 포함한다. 특정 실시형태에서, 상기 화합물은 당이다. 전술한 실시형태와 결합할 수 있는 특정 실시형태에서, 상기 당은 오탄당이다. 전술한 실시형태와 결합할 수 있는 특정 실시형태에서, 상기 당은 육탄당이다. 전술한 실시형태와 결합할 수 있는 특정 실시형태에서, 상기 당은 이당류(disaccharide)이다. 전술한 실시형태와 결합할 수 있는 특정 실시형태에서, 상기 당은 올리고당(oligosaccharide)이다. 다른 실시형태에서, 상기 화합물은 식물 페놀(plant phenol)이다. 전술한 실시형태와 결합할 수 있는 특정 실시형태에서, 상기 식물 페놀은 퀸산(quinic acid)이다. 전술한 실시형태와 결합할 수 있는 특정 실시형태에서, 상기 식물 페놀은 니코틴아미드(nicotinamide)이다. 다른 실시형태에서, 상기 화합물은 피루브산염(pyruvate) 또는 유산염(lactate)이다.
또 다른 양상은, 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하되, 상기 폴리뉴클레오티드는, 내용이 본원의 참조문헌으로 인용된 Supplemental Data, Dataset SI, page 3 in Tian et al ., PNAS , 2009, vol. 106, no. 52, 22157- 22162, 표 10, 표 15 에 나타낸 네우로스포라 또는 피치아 스티피티스 유전자 중 어떠한 것, 또는 NCU01517, NCU09133 또는 NCU10040 에 의해 코드되는 폴리펩티드를 코드하며, 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 더 빠른 비율로 성장하는, 바이오매스 중합체를 포함하는 배지에서 세포를 배양하는 것을 포함하는 바이오매스 중합체에서 세포의 성장을 증가시키는 방법으로 포함한다. 특정 실시형태에서, 상기 폴리뉴클레오티드는, 서열 NCU00130.2, NCU00248.2, NCU00326.2, NCU00762.2, NCU00810.2, NCU00890.2, NCU03328.2, NCU03415.2, NCU03731.2, NCU03753.2, NCU04197.2, NCU04249.2, NCU04287.2, NCU04349.2, NCU04475.2, NCU04997.2, NCU05057.2, NCU05159.2, NCU05493.2, NCU05751.2, NCU05770.2, NCU05932.2, NCU06009.2, NCU06490.2, NCU07340.2, NCU07853.2, NCU07997.2, NCU08744.2, NCU08746.2, NCU08760.2, NCU09108.2, NCU09495.2, NCU09680.2 또는 NCU10045.2 중 어떠한 것을 코드되는 폴리펩티를 코드한다. 특정 실시형태에서, 상기 폴리뉴클레오티드는 NCU07705 에 의해 코드되는 폴리펩티드를 코드한다. 특정 실시형태에서, 상기 재조합 폴리뉴클레오티드는, 표 10, Supplemental Data, Dataset SI, page 3 in Tian et al ., 2009, 또는 표 15 에 나타낸 네우로스포라 또는 피치아 스티피티스 유전자 중 어떠한 것을 코드하는 폴리펩티드와 적어도 50 % 의 아미노산 동일성을 갖는 폴리펩티드를 코드한다. 특정 실시형태에서, 상기 폴리뉴클레오티드는, 서열 NCU00130.2, NCU00248.2, NCU00326.2, NCU00762.2, NCU00810.2, NCU00890.2, NCU03328.2, NCU03415.2, NCU03731.2, NCU03753.2, NCU04197.2, NCU04249.2, NCU04287.2, NCU04349.2, NCU04475.2, NCU04997.2, NCU05057.2, NCU05159.2, NCU05493.2, NCU05751.2, NCU05770.2, NCU05932.2, NCU06009.2, NCU06490.2, NCU07340.2, NCU07853.2, NCU07997.2, NCU08744.2, NCU08746.2, NCU08760.2, NCU09108.2, NCU09495.2, NCU09680.2 또는 NCU10045.2 중 어떠한 것에 의해 코드되는 폴리펩티드와 적어도 50 % 의 아미노산 동일성을 갖는 폴리펩티드를 코드한다. 특정 실시형태에서, 상기 재조합 폴리뉴클레오티드는 NCU07705 에 의해 코드되는 폴리펩티드와 적어도 50 % 의 아미노산 동일성을 갖는 폴리펩티드를 코드한다. 특정 실시형태에서, 상기 바이오매스 중합체는 셀룰로스이다. 다른 실시형태에서, 상기 바이오매스 중합체는 헤미셀룰로스이다. 특정 실시형태에서, 숙주 세포는, 사카로미세스 세레비시아 ( Saccharomyces cerevisiae ), 대장균( Escherichia coli ), 자이모나스 모빌리스( Zymomonas mobilis ), 네우로스포라 크라사( Neurospora crassa ), 칸디다 쉬하테 ( Candida shehatae ), 클로스트리듐( Clostridium sp .), 클로스트리듐 파이토퍼멘탄스( Clostridium phytofermentans), 클로스트리듐 써모셀럼 ( Clostridium thermocellum ), 무렐라 서모아세티카( Moorella thermoacetica ), 서모안아에로박테리움 사카로락티쿰( Thermoanaerobacterium saccharolyticum ), 클레브시엘라 옥시토카(Klebsiella oxytoca) 및 피치아 스티피티스(Pichia stipitis)으로 이루어진 군으로부터 선택된 것이다. 특정 실시형태에서, 숙주 세포는 재조합 폴리뉴클레오티드에 작동가능하게 연결된 유도성 프로모터를 더 포함한다. 특정 실시형태에서, 셀룰라아제의 발현은 재조합 폴리뉴클레오티드의 발현으로 숙주 세포에서 증가되었다. 다른 실시형태에서, 헤미셀룰라아제의 발현은 재조합 폴리뉴클레오티드의 발현으로 숙주 세포에서 증가되었다.
또 다른 양상은, 내인성 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하되, 상기 폴리뉴클레오티드는, 표 10, 내용이 본원의 참조문헌으로 인용된 Supplemental Data, Dataset SI, page 3 in Tian et al ., PNAS , 2009, vol. 106, no. 52, 22157- 22162, 또는 표 15 에 나타낸 네우로스포라 또는 피치아 스티피티스 유전자 중 어떠한 것에 의해 코드되는 폴리펩티드를 코드하며, 또는 내인성 폴리뉴클레오티드의 발현을 저해하고, 및 숙주 세포는 내인성 폴리뉴클레오티드의 발현을 저해하지 않는 세포보다, 배지에서 더 빠른 비율로 성장하는, 바이오매스 중합체를 포함하는 배지에서 세포를 배양하는 것을 포함하는, 바이오매스 중합체에서 세포의 성장을 증가시키는 방법을 포함한다. 특정 실시형태에서, 상기 내인성 폴리뉴클레오티드는 서열 NCU00130.2, NCU00248.2, NCU00326.2, NCU00762.2, NCU00810.2, NCU00890.2, NCU03328.2, NCU03415.2, NCU03731.2, NCU03753.2, NCU04197.2, NCU04249.2, NCU04287.2, NCU04349.2, NCU04475.2, NCU04997.2, NCU05057.2, NCU05159.2, NCU05493.2, NCU05751.2, NCU05770.2, NCU05932.2, NCU06009.2, NCU06490.2, NCU07340.2, NCU07853.2, NCU07997.2, NCU08744.2, NCU08746.2, NCU08760.2, NCU09108.2, NCU09495.2, NCU09680.2 또는 NCU10045.2 의 어떠한 것에 의해 코드되는 폴리펩티드를 코드한다. 특정 실시형태에서, 상기 내인성 폴리뉴클레오티드는 NCU05137 에 의해 코드되는 폴리펩티드를 코드한다. 특정 실시형태에서, 상기 내인성 폴리뉴클레오티드는, 표 10, Supplemental Data, Dataset SI, page 3 in Tian et al ., 2009 또는 표 15 에 나타낸 네우로스포라 또는 피 치아 스티피티스 유전자 중 어떠한 것에 의해 코드되는 폴리펩티드와 적어도 50 % 의 아미노산 동일성 동일성을 갖는 폴리펩티드를 코드한다. 특정 실시형태에서, 상기 내인성 폴리뉴클레오티드는, 서열 NCU00130.2, NCU00248.2, NCU00326.2, NCU00762.2, NCU00810.2, NCU00890.2, NCU03328.2, NCU03415.2, NCU03731.2, NCU03753.2, NCU04197.2, NCU04249.2, NCU04287.2, NCU04349.2, NCU04475.2, NCU04997.2, NCU05057.2, NCU05159.2, NCU05493.2, NCU05751.2, NCU05770.2, NCU05932.2, NCU06009.2, NCU06490.2, NCU07340.2, NCU07853.2, NCU07997.2, NCU08744.2, NCU08746.2, NCU08760.2, NCU09108.2, NCU09495.2, NCU09680.2, 또는 NCU10045.2 의 어떠한 것에 의해 코드되는 폴리펩티드와 적어도 50 % 동일한 아이노산을 갖는 폴리펩티드를 코드한다. 특정 실시형태에서, 상기 내인성 폴리뉴클레오티드는 NCU05137 에 의해 코드되는 폴리펩티드와 적어도 50 % 동일한 아이노산을 갖는 폴리펩티드를 코드한다. 특정 실시형태에서, 숙주 세포는, 사카로미세스 세레비시아( Saccharomyces cerevisiae ), 대장균( Escherichia coli ), 자이모나스 모빌리스( Zymomonas mobilis ), 네우로스포라 크라사 ( Neurospora crassa ), 칸디다 쉬하테( Candida shehatae ), 클로스트리듐( Clostridium sp .), 클로스트리듐 파이토퍼멘탄스( Clostridium phytofermentans ), 클로스트리듐 써모셀럼( Clostridium thermocellum), 무렐라 서모아세티카 ( Moorella thermoacetica ), 서모안아에로박테리움 사카로락티쿰 ( Thermoanaerobacterium saccharolyticum ), 클레브시엘라 옥시토카(Klebsiella oxytoca) 및 피치아 스티피티스(Pichia stipitis)로 이루어진 군으로부터 선택된 것이다. 특정 실시형태에서, 상기 바이오매스 중합체는 셀룰로스이다. 다른 실시형태에서, 바이오매스 중합체는 헤미셀룰로스이다. 특정 실시형태에서, 숙주 세포의 셀룰라아제 활성도는 내인성 폴리뉴클레오티드의 발현의 저해로 증가되었다. 다른 실시형태에서, 숙주 세포의 헤미셀룰라아제 활성도는 내인성 폴리뉴클레오티드의 발현의 저해로 증가되었다. 특정 실시형태에서, 내인성 폴리뉴클레오티드의 발현의 저해는 내인성 폴리뉴클레오티드를 포함하는 유전자의 돌연변이(mutating) 또는 결실(deleting)을 포함한다.
도 1 은 NCU07705 에 의해 코드되는 폴리펩티드의 도메인 구조를 나타낸다.
도 2 는 NCU05137 의 계통분석(phylogenetic analysis)을 나타낸다. 엔. 크라사 NCU05137 의 예측된 오솔로그(predicted orthologs)는 BLAST 에 의한 중요한 유사성을 나타내는 아미노산 서열을 기초로 한 NCBI 및 JGI 로부터 검색하였다. 모든 확인된 실모양의 진균의 오솔로그(filamentous fungal orthologs)를 나타낸다 ; NCBI E 수치는 9e-175 인 B. 푸켈리아나(B. fuckeliana)를 제외한 0.0 이다. NCU05137 의 동족체(Homologs)는 수많은 박테리아에서 또한 확인된다(E 값 ~e-30). 폴라로모나스 나프탈렌이보란스(Polaromonas naphthalenivorans)(베타-프로테오박테리움, beta-proteobacterium)로부터의 YP 981875 는 외집단(outgroup)으로서 사용되었다. A. = 아스페르길루스(Aspergillus) ; N. = 네오사토리아(Neosartorya) ; P. 키로소게늄(P. chyrosogenum) = 페니실리움(Penicillium) ; S. = 스클레오티니아(Sclerotinia) ; B. = 보트리요티니아(Botryotinia) ; P. = 피레노포라(Pyrenophora) ; C. = 코칠레오볼루스(Cochliobolus) ; N. 하마토코카(N. haematococca) = 넥트리아(Nectria) ; P. 안세리나(P. anserina) = 포도스포라(Podospora) ; N. = 네우로스포라(Neurospora). 나무형(tree)은 MEGA3, NJ 에 의해 생성되었다. 아미노산 사이트 당 바(bar) = 0.2 치환(substitutions).
도 3 은 단일 탄소원(sole carbon source)으로서의 미스칸터스 ( Miscanthus ) 및 아비셀(Avicel)에서 성장하였을 때의 N. 크라사 FGSC2489 및 T. 레세이(T. reesei) QM9414 엔도글루카나제 활성도(endoglucanase activity)의 분석을 나타낸 것이다. N. 크라사 WT 계통(strain) FGSC2489 및 T. 레세이(T. reesei) QM9414 의 배양 여과물에서 엔도글루카나제 활성도. N. 크라사는 25 ℃ 에서 단일 탄소원으로서의 2 % 의 아비셀 또는 억세 파우더를 함유하는 Vogel's 최소배지에서 성장하였다. T. 레세이 계통은 25 ℃ 에서 단일 탄소원으로서의 1 % 의 아비셀 또는 억세 파우더를 갖는 MA 배지에서 접종하였다(inoculate). 둘 다의 계통을 동일한 양의 분생자(conidia)[lOO mL 배양물(culture)에 1 x 106 /mL]로 접종하였다. 배양물에서 상이한 시점에서의 엔도글루카나제 활성도를, 제조자의 지시(manufacturer's instructions)(아일랜드에 위치한 Megazyme)에 따라 기질(substrate)로서 Azo-CM-셀룰로스를 사용하여 pH 4.5 에서 측정하였다.
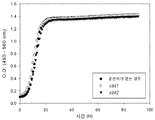
도 4 는 미스칸터스 및 아비셀에서 성장한 N. 크라사의 전사 프로파일링(transcriptional profiling)을 나타낸 것이다. (A) 미스칸터스 배양에서 발현의 차이점으로 나타내는 769 유전자의 계층적 군집 분석(Hierarchical clustering analysis). 어두운 셰이딩(Dark shading)은 상대적으로 보다 높은 발현(expression)을 나타내고, 밝은 셰이딩(light shading)은 상대적으로 낮은 발현을 나타낸다. 라인 1(Lane 1) : 16 시간 동안의 Vogel's 최소 배지 N. 크라사 배양물(Vogel 1956)의 발현 프로파일(Expression profile). 라인 2 : 16 시간 동안의 단일 탄소원으로서의 미스칸터스에서 성장한 배양물의 발현 프로파일. 라인 3, 4, 5 : 40 시간, 5 일 및 10 일 동안의 미스칸터스에서 성장한 배양물로부터의 발현 프로파일. 세 가지 군집(clusters)을 C1, C2 및 C3 으로 나타내었다. 대부분의 셀룰라아제 및 헤미셀룰라아제 유전자의 증가된 발현 수준을 나타낸 군집은 상자 모양을 나타내었다(boxed)(C3 군집). (B) N. 크라사 미스칸터스 대 아비셀 성장한 배양물 사이의 발현 프로파일에서 공통부분(overlap)의 분석. 단백질의 분석 및 공통부분은 이중 질량 분석기(tandem mass spectrometry)(Bottom)로 미스칸터스 및 아비셀에서 성장한 N. 크라사의 배양 여과물에서 측정하였다. (C) 미스칸터스 배양물에서 상대적인 발현 수준에서 증가됨을 나타낸 231 유전자의 기능범주(Functional category, FunCat) 다양한 분석(enrichment analysis)(Ruepp 2004). 분류되지 않은 그룹(the unclassified group)을 포함한 중요한 풍부함(enrichment)(p<0.001)을 나타낸 기능범주를 나타낸다.
도 5 는 최소배지(MM)에서 성장하는 동안 및 16 시간(16 hr), 40 시간(40 hr), 5 일(5 days) 및 10 일(10 days) 동안 미스칸터스(Miscanthus)에서 성장하는 동안의 셀룰라아제(A) 및 헤미셀룰라아제(B)를 코딩하는 N. 크라사 유전자의 상대적인 발현 수준을 나타낸다.
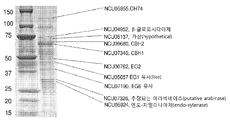
도 6 은 MS 에 의해 확인된 분비된 단백질을 코딩하는 유전자의 삭제를 포함하는 스트레인(strains)으로부터의 배양 상청액(culture supernatants)의 단백질 프로파일(protein profile) 및 효소 활성도(enzymatic activity)를 나타낸 것이다. (A) 7 일 동안 아비셀에서 성장한 야생형(wild type)과 비교한 16 결실 스트레인(deletion strains)의 배양 여과물(culture filtrates)에 나타난 단백질의 SDS-PAGE. 삭제 스트레인(strain)은 미스칸터스 및 아비셀 배양 여과물 둘 다에서의 질량 분석법(mass spectrometry)에 의한 단백질의 식별(identification)을 기반으로 선택되었다. 스트레인은 유전자 NCU 번호를 기초로 정리하였고, 야생형 스트레인(wild-type strain)은 FGSC 2489 이다. 삭제된 유전자에 해당하는 누락된 단백질 밴드(Missing protein bands)는 상자모양으로 나타났다(boxed). (B) 총 분비된 단백질, 아조-CMCase(azo-CMCase) 및 β-글루코시다아제 활성도 검정(실시예 5 참고)을 (A)로부터의 동일한 샘플을 사용하여 16 삭제 스트레인 및 야생형 부모 스트레인(wild-type parental strain)(FGSC 2489)에서 실행하였다. 활성도 및 단백질 농도를 야생형 농도과 비교하여 정규화시키고, 3 번의 생물학적인 측정(triplicate biological measurements)의 평균을 나타내었다. (C) (A) 에서의 동일한 샘플을 사용한 16 삭제 스트레인로부터의 배양 여과물의 셀룰라아제 활성도. 아비셀라아제 활성도(Avicelase activity)를 측정하기 위해 배양 여과물(Culture filtrates) 10 배 희석하였고 5 mg/mL 아비셀과 혼합하였다(실시예 5 참고). 40 ℃ 에서 배양하여 8 시간 후에 글루코스(흑색) 및 셀로비오스(cellobiose)(백색)를 측정하였다.
도 7 은 단일 탄소원으로서 아비셀에서 성장한 배양물로부터의 돌연변이체 분석을 기반으로 한 N. 크라사 분비된 단백질의 정체(identity)를 나타낸 것이다. 25 ℃ 에서 7 일 동안 100 mL 의 진탕 플라스크(shake flasks)에서 2 % 아비셀에서 성장한 WT N. 크라사(FGSC 2489)로부터의 분비된 단백질의 SDS-PAGE. 15 μL 의 농축시키지 않는 배양 여과물을 크라이테리언 4-15 % 26-웰 겔 내에 로딩하였다(loaded). 내셔널 진단(National Diagnostics)으로부터는 포토 블루 세이프(Proto Blue Safe)(Coomassie)를 겔을 착색시키는데(stain) 사용하였다. 단백질 밴드를, 결실 스트레인(deletion strains)에서 분비된 단백질의 분석을 기반으로 한 도 6A 에 나타낸 바와 같은 이러한 연구에서 확인하였다.
도 8 은 △NCU04952 및 △ANCU05137 에서 cbh-1(NCU07340) 및 gh6-2 (NCU09680; CBHII)의 발현 및 분비된 단백질 프로파일을 나타낸 것이다. (A) WT, △NCU04952, 및 △NCU05137 에서 총 분비된 단백질의 SDS-PAGE. 배양물을 분생자(conidia)로부터의 아비셀에서 배양물을 성장시키고 30 시간, 2 일(48 시간) 및 3 일(72 시간)에 채취하였다(harvested)(실시예 5 참고). 라인 1-3 은 각각의 WT, △NCU04952 및 △NCU05137 스트레인(strains)로부터의 아비셀에서 30 시간 성장 후의 20x 농축시킨 배양 여과물이다. 라인 4-6 은 각각의 WT, △NCU04952 및 △NCU05137 스트레인로부터의 아비셀에서 2 일 성장 후의 농축시키지 않은 배양 여과물이다. 라인 7-9 는 각각의 WT, △NCU04952 및 △NCU05137 스트레인으로부터의 3 일후의 농축시키지 않은 배양 여과물이다. (B) 아비셀에서 성장하는 동안의 WT, △NCU04952 및 △NCU05137 스트레인에서 cbh -1(NCU07340; CBHI) 및 gh6-2(NCU09680; CBHII)의 RT-PCR. WT 및 삭제 스트레인은 분생자로부터의 아비셀에서 성장하였고, 48 시간 및 72 시간에 채취하였다(실시예 5 참고). 단일 탄소원으로서의 수크로스를 갖는 최소배지(MM) 배양물(Vogel 1956)을 16 시간 동안 성장시켰다[유사한 발달 시점(developmental time point)]. cbh -1 및 gh6-2 의 폴드 인덕션(fold induction)은, 모든 샘플의 대조군으로서 사용된 액틴 유전자 발현(actin gene expression)을 갖는 MM 조건 하에서 이러한 유전자의 발현과 관련되어 있다.
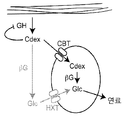
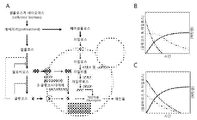
도 9 는 N. 크라사에서 식물 세포 벽 디컨스트럭션(deconstruction)의 모델을 나타낸 것이다. 도입(induction) : 낮은 수준에서 발현된 세포외 효소(Extracellular enzymes)는 대부분 분비된 식물 세포 벽 분해효소(plant cell wall degrading enzymes)를 코딩하는 유전자의 발현 수준을 극적으로 증가시키기 위해 단일 N. 크라사의 2 차 대사물질(secondary metabolites)를 생성한다. 이용(utilization) : 세포 벽 분해산물(cell wall degradation products)의 전좌(translocation)에 특이적인 세포외 효소 및 운반체(transporters)는 N. 크라사가 성장에 대한 식물 세포 물질을 활용할 수 있게 한다. 몇몇의 세포외 단백질(NCU05137, NCU05057, 및 NCU04952)은 활용 단계에서 셀룰라아제(cellulases) 및 헤미셀룰라아제(hemicellulase )의 유전자 발현을 조절하는 대사물질(metabolites)을 생성할 수 있다 ; 이중의 육각형(double hexagon)(셀로비오스), 이중의 오각형(double pentagon)(자이로비오스, xylobiose), 육각형(글루코스) 및 오각형(자일로스). 그려진 식물 세포 벽-분해 효소는 CBH(I), CBH(II), EG2, EG1, EG6 및 자일라나아제(xylanase)를 포함한다. 추가적인 셀룰로이스 분해효소(cellulolytic enzymes)를 나타내지 않는다. 화살표의 두께는 반응의 상대적인 강도를 나타낸다.
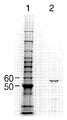
도 10 은 S. 서머파일 단백질 서열(S. thermophile protein sequences)의 데이터베이스에 대한 N. 크라사 추정상의 운반체(putative transporters)의 서열 또는 N. 크라사 단백질 서열의 데이터베이스에 대한 S. 호열성 추정상의 운반체의 서열을 BLAST 에서 검색한 결과를 나타낸 것이다.
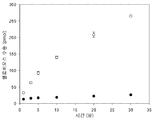
도 11 은 NCU08114 가 결핍된 N. 크라사 스트레인의 성장 표현형(growth phenotype)을 나타낸 것이다. (A) 탄소원으로서 결정질 셀룰로스(crystalline cellulose)를 갖는 성장의 3 일 후의 WT(왼쪽) 및 △NCU08114(오른쪽) N. 크라사 스트레인의 진탕 플라스크(Shaker flasks). (B) 각각의, 16 시간 또는 28 시간 동안, 탄소원으로서 수크로스 또는 결정질 셀룰로스을 이용하여 성장한 N. 크라사 배양물로부터의 평균 알라마 블루ⓒ 형광(Mean Alamar Blueⓒ fluorescence). 형광을 100 % 로 WT 에 맞추어 정규화시켰다(normalize). 에러 바(Error bars)를 세 번의 생물학적 반복(biological replicates)으로부터의 측정(measurements) 사이의 표준 편차(standard deviation)이다. NCU00801이 결핍된 N. 크라사는, 단당류 운반체에 의해 순차적으로 계속되는 셀로덱스트린에서 글루코스로 가수분해하는(Scarborough 1973) N. 크라사 분비된 β-글루코시다아제의 명백한 표현형을 갖지 않는다(Tian 등의, 2009). 이러한 소비(consumption)의 우회 루트(alternate route)는 이러한 삭제 라인(deletion lines)에서 셀로덱스트린 운반체의 결함의 과소평가(underestimate)로 이어진다.
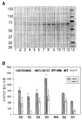
도 12 는 (A) NCU00130 와 함께 NCU00801, NCU05853 또는 NCU08114 가 발현된 S. 세레비시아 스트레인(S. cerevisiae strains)에 대한 셀로비오스(cellobiose)의 소비 ; (B) NCU00130 와 함께 NCU00801, NCU05853 또는 NCU08114 가 발현된 S. 세레비시아 스트레인에 대한 셀로트리오스(cellotriose)의 소비 ; (C) NCU00130 와 함께 NCU00801, NCU05853 또는 NCU08114 가 발현된 S. 세레비시아 스트레인에 대한 셀로테트라오스(cellotetraose)의 소비 ; 및 (D) NCU00130 와 함께 NCU00801, NCU05853 또는 NCU08114 가 발현된 S. 세레비시아 스트레인에 대한 셀로헥사오스(cellohexaose)의 소비를 나타낸 것이다.
도 13 은 NCU008114 또는 NCU00801 이 결핍된 N. 크라사 스트레인에 의한 셀로덱스크린의 소비를 나타낸 것이다. 나타낸 N. 크라사 스트레인을 15 분 동안 90 μL 의 각각의 당으로 배양하였다. 바(Bars)는 두 개의 독립적인 실험으로부터의 배양 후에 상청액에 남아있는 당의 평균 농도를 나타낸 것이다. 에러 바는 이들 실험 사이의 표준 편차이다.
도 14 는 NCU00801/cbt1 이 발현된 S. 세레비시아 스트레인(S. cerevisiae strain)에 의한 셀로비오스 수송을 나타낸 것이다. CBT 1 이 있을 때(○) 또는 없을 때(●)에 의한 셀로비오스 수송을 나타낸 것이다. 스트레인 둘 다는 세포 내의 β-글루코시다아제, NCU00130 가 발현되었다. 셀로비오스의 초기 농도는 50 μM 이다. 모든 값은, 이러한 측정 사이의 표준 편차를 나타낸 에러 바와 함께 두 측정 사이의 평균이다.
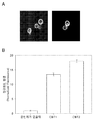
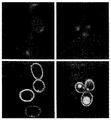
도 15 는 CBTl 및 CBT2 에 융합시킨(fused) GFP 의 위치(localization) 및 정량(quantification)을 나타낸 것이다. (A) 이들의 C-말단에 GFP 에 융합시킨 cbtl(왼쪽) 또는 cbt2(오른쪽)에 발현된 S. 세레비시아 스트레인의 이미지. (B) 셀로비오스 운반체가 없는 또는 이들의 C-말단에 GFP 에 융합시킨 cbtl 또는 cbt2 가 발현된 효모 스트레인의 GFP 형광(fluorescence). 값은 세 번의 생물학적 반복으로부터의 평균이고, 에러 바는 이들의 반복 사이의 표준 편차를 나타낸다.
도 16 은 S. 세레비시아에서 발현된 N. 크라사 수송 시스템에 의한 셀로덱스트린 수송을 나타낸 것이다. (A) 유전자 NCU00801(cbtl 으로 명명, ○), NCU08114 (cbt2 로 명명, ▼)가 발현되거나, 또는 운반체가 없는 경우(●)의 효모 스트레인의 셀로비오스-매개 성장(Cellobiose-mediated growth). 모든 스트레인은 또한 세포 내의 β-글루코시다아제, NCU00130 를 발현하였다. 대표적인 실험을 나타내었다. 세 가지의 독립적인 실험으로부터의 성장비율을 하기와 같다 : cbtl, 0.0341 ± 0.0010 hr-1; cbt2, 0.0131 ± 0.0008 hr-1 ; 운반체가 없는 경우, 0.0026 + 0.0001 hr-1. (B) 셀로트리오스 및 셀로테트라오스에서 효모 스트레인의 성장. 범례(legend)에 열거된 운반체 뿐만 아니라 세포내의 β-글루코시다아제, NCU00130 가 발현된 스트레인은 단일 탄소원으로서 제공된 0.5% (w/v)의 셀로트리오스(G3) 또는 셀로테트라오스(G4)와 함께 성장시켰다. 대표적인 실험을 나타내었다. 세 가지 독립적인 실험으로부터의 성장율(Growth rates)은 하기와 같다 : cbtl 셀로트리오스, 0.0332 + 0.0004 hr-1; cbtl 셀로테트라오스 0.0263 + 0.0020 hr-1 ; 운반체가 없는 셀로트리오스, 0.0043 + 0.0015 hr-1; cbt2 셀로트리오스, 0.0178 + 0.0005 hr-1; cbt2 셀로테트라오스 0.0041 + 0.0003 hr-1; 운반체가 없는 셀로테트라오스, 0.0031 + 0.0008 hr-1. (C) 글루코스는 정제된 NCU00130 에 의해 셀로비오스(G2), 셀로트리오스(G3) 및 셀로테트라오스(G4) 가수분해로부터 생산되었다. 세 가지의 독립적인 측정의 평균 및 표준 편차를 나타내었다. 효소(2 nmol)가 없는 배양에서의 잔류 글루코스는 나타낸 값으로부터 뺐다.
도 17 은 글루코스에서 운반체가 없거나(●) 또는 cbt1(○), cbt2(▼)가 발현된 S. 세레비시아 스트레인의 성장을 나타낸 것이다. 모든 스트레인은 β-글루코시다아제, NCU00130 이 발현되었다. 대표적인 실험을 나타내었다.
도 18 은 250 mL 의 플라스크에서 S. 세레비시아 스트레인의 셀로비오스-매개 성장을 나타낸 것이다. 값은 β-글루코시다아제, NCU00130, cbtl 또는 cbt2 가 발현된 효모 스트레인, 또는 어떠한 운반체도 결핍된 스트레인 발현 NCU00130 의 두 번의 복제 배양물(two replicate cultures) 사이의 평균 OD 를 나타낸 것이다. 에러 바는 복제 사이의 표준 편차를 나타낸다.
도 19 는 CBT1 및 CBT2 에 의한 셀로비오스 수송의 반응속도(kinetics)를 나타낸 것이다. 셀로비오스 수송의 비율은 cbtl 또는 cbt2 가 발현된 효모 스트레인에 의한 셀로비오스 농도의 작용으로서 측정하였다. 수송 비율(transport rate)은 풍부한 운반체(transporter abundance)에 대해서 정규화시켰다(normalize).
도 20 은, 셀로비오스, 셀로트리오스 또는 셀로테트라오스에서 성장한 x-축에 나타낸 네우라스포라 유전자(Neurospora genes)의 조합을 발현된 S. 세레비시아의 능력을 나타낸 것이다.
도 21 은 cbtl 또는 cbt2 를 운반하는 스트레인에서 셀로비오스 수송을 위한 셀로덱스트린의 경쟁(competition)을 나타낸 것이다. 각각의 분류되지 않은 당(unlabeled sugar)의 5-폴드 초과량(5-fold excess)은 [3H]-셀로비오스 수송의 검정 동안에 포함되었다. CBT1 또는 CBT2 의 기질(Substrate)은 결합을 위한 경쟁에 의해 [3H]-셀로비오스 수송 비율을 감소시켰다. 바는 세 번의 반복으로부터의 평균을 나타낸다. 에러 바는 이러한 반복 사이의 표준 편차를 나타낸다. 값은 100 까지의 경쟁 당 없이 [3H]-셀로비오스 수송 비율에 맞추어 정규화시켰다.
도 22 는 정제된 NCU00130 의 SDS-PAGE 겔을 나타낸 것이다. 라인 1, kDa 에서의 단백질 분자량 기준. 라인 2, 니켈-NTA 레진(nickel-NTA resin) 상에서 여과한 후의 NCU00130. kDa 의 분자량은 왼쪽에 나타내었다.
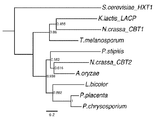
도 23 은 셀로비오스 운반체 NCU08114 및 NCU00801 의 최고 가능성을 갖는 계통발생론적 분석(maximum likelihood phylogenetic analysis)을 나타낸 것이다. S. 세레비시아 HXT1 및 K. 락티스 LACP 를 예외로 하는, 나타낸 단백질을 코딩하는 모든 유전자는, 균류(fungus)가 식물 세포 벽 물질 또는 셀로비오스와 접촉하여 쓰이기 시작할 때, 발현 수준을 증가시키기 위한 것으로 알려졌다(Tian 등의, 2009; Noguchi 등의, 2009; Wymelenberg 등의, 2010; Martin 등의, 2010). S. 세레비시아 HTX1, 낮은 친화도(low affinity)의 글루코스 운반체(Reifenberger 등의, 1997)는 외집단(outgroup)으로서 사용되었다.
도 24 는 셀로비오스의 발효(fermentation), N. 크라사로부터의 셀로비오스 수송 시스템이 발현된 S. 세레비시아에 의한 동시의 당화(saccharification) 및 셀룰로스의 발효를 나타낸 것이다. (A) 에탄올에 대한 셀로비오스 발효. 에탄올은 CBTl 을 갖거나(●) 또는 CBTl 을 갖지 않은(○) 효모 스트레인에 의해 생산되었다. CBTl 을 갖거나(▼) 또는 CBTl 을 갖지 않은(△) 효모 스트레인을 사용한 발효 반응 동안에 셀로비오스 농도. (B) CBTl 을 갖거나, 갖지 않은 효모 스트레인을 사용한 SSF. CBTl 을 갖는 스트레인의 존재 하에서 셀로비오스(●) 및 글루코스(▼)의 농도, 및 CBTl 이 결핍된 스트레인의 존재 하에서 셀로비오스(○) 및 글루코스(△)의 농도. 참고로 0.1 mg/mL 셀로비오스 = 292 μM. (C) CBTl 을 갖는(●) 또는 CBTl 을 갖지 않는(○) 스트레인을 사용한 SSF 동안 생산된 에탄올. 모든 패널(panels)에서, 값은 세 번의 생물학적인 반복의 평균이다. 에러 바는 이러한 반복 사이의 표준 편차이다. 모든 스트레인은 세포 내의 β-글루코시다아제, NCU00130 을 발현시켰다.
도 25 는 효모에 의한 셀롤로스의 동시의 당화(simultaneous saccharification) 및 발효 동안에 곰팡이(filamentous fungi)로부터의 셀로덱스트린 수송 경로(transport pathways)의 사용을 나타낸 것이다. 셀로덱스트린(Cdes) 수송 경로(흑색)은 셀로덱스트린 운반체(CBT) 및 세포 내의 β-글루코시다아제(βG)를 포함한다. 표준 효모(standard yeast)에 나타낸 당 이화 경로(sugar catabolism pathway)는 육탄당 운반체(hexose transporters, HXT)를 포함한다. SSF 에서 셀룰라아제(GH) 및 세포외 β-글루코시다아제(βG) 둘 다는 사용될 수 있다.
도 26 은 기능적으로 중요한 NCU00801 및 NCU0811 에서의 잔여물(residues)을 나타낸 것이다. (A) cbtl/NCU00801 의 Ala-스캔(Ala-scan). (B) cbtl NCU00801 의 폴리펩티드 서열[표시된 중요한 잔기(important residues marked)]. (C) cbt2/NCU08l14 의 폴리펩티드 서열(표시된 중요한 잔기).
도 27 은 P. 스티피티스(P. stipitis)로부터의 셀로비오스 운반체가 발현된 S. 세레비시아 스트레인의 비교를 나타낸 것이다. (A) β-글루코시다아제가 발현된 S. 세레비시아 스트레인 및 셀로비오스 운반체 NCU00801, NCU08114 및 NCU05853 의 오솔로그(orthologs)의 세포 성장. (B) P. 스티피티스로부터의 셀로비오스 운반체의 비교 : β-글루코시다아제 및 셀로비오스 운반체가 발현된 S. 세레비시아 스트레인의 세포 성장. (C) P. 스티피티스로부터의 셀로비오스 운반체의 비교 : β-글루코시다아제 및 셀로비오스 운반체가 발현된 S. 세레비시아 스트레인에 의한 자일로스 소비 및 에탄올 생산.
도 28 은 셀로비오스 운반체 오솔로그의 배열을 나타낸 것이다. (A) 테스트된 조건 하에서 운반체 기능을 갖는 것으로 나타나지 않은 하나를 포함하는 셀로비오스 운반체 오솔로그의 배열. (B) 운반 기능을 갖는 셀로비오스 운반체 오솔로그의 배열. (C) NCU00801 및 NCU08114 의 배열.
도 29 는 NCU00801 및 NCU08114 의 상동성 모델(homology models)에서 표시된 기능적으로 중요한 모티프(motifs)를 나타낸 것이다. (A) NCU00801 의 상동성 모델에서 셀로비오스 운반체 모티프의 위치. 모티프는 흑색으로 강조되었다. 모티프 [LIVM]-Y-[FL]-x(13)-[YF]-D 를 페널(panels) a 및 b 에 나타내었다. 모티프 [YF]-x(2)-G-x(5)-[PVF]-x(6)-[DQ]를 페널 c 및 d 에 나타내었다. 모티프 G-R-[RK] 를 페널 e 및 f 에 나타내었다. 모티프 R-x(6)-[YF]-N 를 페널 g 및 h 에 나타내었다. 모티프 WR-[IVLA]-P-x(3)-Q 를 페널 i 및 j 에 나타내었다. 모티프 P-E-S-P-R-x-L-x(8)-A-x(3)-x(2)-Y-H 를 페널 k 및 l 에 나타내었다. 모티프 F-[GST]-Q-x-S-G-N-x-[LIV]를 페널 m 및 n 에 나타내었다. 모티프 L-x(3)-[YIV]-x(2)-E-x-L-x(4)-R-[GA]-K-G 를 페널 o 및 p 에 나타내었다. 참고 : 이러한 이미지에서, 트랜스멤브레인 헬릭스 6 및 7 을 연결하는 몇몇의 잔기는, 그들이 구멍을 가렸기 때문에(as they occlude the pore) 명확하게 제거되었다. (B) NCUQ8114 상동성 모델에서 셀로비오스 운반체 모티프의 위치. 모티프는 흑색으로 강조되었다. 모티프 [LIVM]-Y-[FL]-x(13)-[YF]-D 를 페널 a 및 b 에 나타내었다. 모티프 [YF]-x(2)-G-x(5)-[PVF]-x(6)-[DQ]를 페널 c 및 d 에 나타내었다. 모티프 G-R-[RK]를 페널 e 및 f 에 나타내었다. 모티프 R-x(6)-[YF]-N 을 페널 g 및 h 에 나타내었다. 모티프 WR-[IVLA]-P-x(3)-Q 를 페널 i 및 j 에 나타내었다. 모티프 P-E-S-P-R-x-L-x(8)-A-x(3)-x(2)-Y-H 를 페널 k 및 l 에 나타내었다. 모티프 F-[GST]-Q-x-S-G-N-x-[LIV]를 페널 m 및 n 에 나타내었다. 모티프 L-x(3)-[YIV]-x(2)-E-x-L-x(4)-R-[GA]-K-G 를 페널 o 및 p 에 나타내었다. 참고 : 이러한 이미지에서, 트랜스멤브레인 헬릭스 6 및 7 을 연결하는 몇몇의 잔기는, 그들이 구멍을 가렸기 때문에 명확하게 제거되었다.
도 30 은 플라스미드 발현의 구성에서 사용된 클로닝 과정(cloning process)을 나타낸 것이다 : (A) 추정상의 운반체(putative transporters) 및 (B) 운반체-GFP 융합 단백질(transporter-GFP fusion proteins).
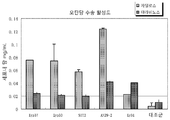
도 31 은 글루코스-흡수 활성도(glucose-uptake activity)를 갖는 것으로 확인된 추정상의 운반체(putative transporters)의 오탄당 수송 활성도를 나타낸 것이다.
도 32 는 글루코스-흡수 활성도(glucose-uptake activity)를 갖지 않는 것으로 확인된 추정상의 운반체의 오탄당 수송 활성도를 나타낸 것이다.
도 33 는 NCU00821(AN25), STL12/XUT6(Xyp29) 및 XUTl(Xyp32)의 오탄당 흡수를 나타낸 것이다. A 는 자일로스 흡수를 나타내고 B 는 아라비노스 흡수를 나타낸 것이다.
도 34 는 STL12 XUT6 가 발현된 S. 세레비시아에 의한 l4C-표지된(labeled) 당 흡수를 나타낸 것이다(Xyp29).
도 35 는 GFP 형광에 의해 관찰된 바와 같은 S. 세레비시아 세포에서 발현된 운반체의 위치(localizations)를 나타낸 것이다. 첫 번째 줄의 왼쪽에서 오른쪽 : NCU00821-GFP 형광, NCU00821 핵(nuclei) ; 두 번째 줄의 왼쪽에서 오른쪽 : STL12 XUT6-GFP 형광, STL12/XUT6 핵.
도 36 은 (A) NCU00821(AN25), (B) STL12/XUT6(Xyp29) 및 (C) XUTl(Xyp32)으로 발현된 완충되지 않은 세포 현탁액에 말토오스의 첨가 상에서 pH 에서의 효과를 나타낸 것이다. 검정색 화살표는 말토오스를 첨가하였을 때의 시점(time points)을 나타낸 것이다.
도 37 은 NCU00821, STL12/XUT6 및 XUT1 의 공수송체 검정(syraporter assay)의 결과를 나타낸 것이다. (A) 자일로스에 대한 NCU00821. (B) 아라비노스에 대한 NCU00821. (C) 아라비노스에 대한 XUTl. (D) 자일로스에 대한 NCU00821. (E) 아라비노스에 대한 STL12 XUT6. (F) 자일로스에 대한 STLl2/XUT6. 검정색 화살표는 말토오스를 첨가하였을 때의 시점을 나타낸 것이다.
도 38 은 운반체의 과잉발현(overexpression)의 표현형 분석(phenotypic analyses)를 나타낸 것이다. (A) 는 OD 를 나타낸 것이고, (B) 는 자일로스 농도를 나타낸 것이고, (C) 는 0.5 % 자일로스-함유 배지에서의 자일로스 소비를 나타낸 것이다. (D) 는 OD 를 나타낸 것이고, (E) 는 자일로스 농도를 나타낸 것이고, (F) 는 0.5 % 자일로스-함유 배지에서의 자일로스 소비를 나타낸 것이다. (G) 는 다복제 플라스미드(multicopy plasmid), pRS424 에서 도입된 오탄당 운반체를 함유하는 S. 세레비시아의 성장 곡선을 나타낸 것이다.
도 39 는 이종 운반체(heterologous transporters)의 클로닝(cloning)에 사용된 플라스미드의 맵(maps)을 나타낸 것이다.
도 40 은 오탄당 운반체 오솔로그가 발현된 S. 세레비시아 스트레인에 의한 당-흡수 검정의 결과를 나타낸 것이다.
도 41 은 Clustal W (1.81)에 의한 오탄당 운반체 오솔로그의 서열 정렬(sequence alignments)을 나타낸 것이다. (A) 자일로스 운반체 오솔로그의 정렬. (B) 아라비노스 운반체의 정렬. (C) 자일로스 및 아라비노스 운반체의 정렬. 컨센서스 키(Consensus key) : * - 단일, 완전히 보존된 잔기(single, fully conserved residue) ; : - 강한 그룹(strong groups)의 보존(conservation) ; - 약한 그룹(weak groups)의 보존.
도 42 는 자일로스-이용 효소(xylose-utilizing enzymes)를 발현시키기 위한 제작한(engineered) 상이한 S. 세레비시아 스트레인을 나타낸 것이다.
도 43 은 자일로스 이용 경로 효소(xylose utilization pathway enzymes)를 함유하는 동일한 카세트(cassettes)가 발현된 상이한 백그라운드(backgrounds)의 세 가지 S. 세레비시아 스트레인의 자일로스 대사(xylose metabolism)(자일로스 소비 및 에탄올 생산 등과 같이 모니터 한 바와 같이)를 나타낸 것이다.
도 44 는 자일로스 이용 경로 효소를 함유하는 동일한 카세트를 발현하는 상이한 백그라운드의 세 가지 S. 세레비시아 스트레인의 자일로스-흡수 비율 및 대사물 수득률(metabolite yield)을 나타낸 것이다.
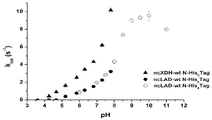
도 45 는 다양한 조건 하에서 S. 세레비시아 스트레인 DA24 에 의한 자일로스 발효(xylose fermentation)를 나타낸 것이다. (A) 진탕 플라스크에서 40 g/L 의 자일로스, (B) 진탕 플라스크에서 80 g/L 의 자일로스 및 (C) 바이오리액터(bioreactor)에서 80 g/L 의 자일로스. 부호 : 자일로스(■), 에탄올(◆) 및 OD600(●).
도 46 은 (A) S. 세레비시아 DA24 및 (B) P. 스티피티스 (P. stipitis ) 사이의 자일로스 소비 및 에탄올 생산의 비교를 나타낸 것이다. 부호 : 자일로스(■), 에탄올(◆) 및 OD600(●).
도 47 은 유전자 조작된(engineered) S. 세레비시아에서의 자일로스 대사 상에서 XYL2 과잉-발현 수준(over-expression levels)를 테스트하는데 사용된 실험 설계(experimental design)를 나타낸 것이다.
도 48 은 유전자 조작된(engineered) 자일로스-발효 S. 세레비시아 내로의 추가적인 XYL2 통합(integration)(즉, 증가된 XYL2 발현 수준)의 효과를 나타낸 것이다.
도 49 는 유전자 조작된 S. 세레비시아 에 의한 자일로스에서 XYL2 및 XYL3추가적인 동시의 과잉-발현(additional simultaneous over-expression)의 효과를 나타낸 것이다.
도 50 은 상이한 수준의 자일로스-발효 효소(xylose-fermenting enzymes)가 발현된 S. 세레비시아 스트레인을 나타낸 것이다.
도 51 은 유전자 조작된 S. 세레비시아 에 의한 차별된 XYL1 발현(differential XYL1 expression)의 효과를 나타낸 것이다.
도 52 는 상이한 환원효소(reductases)를 갖는 동일한 XYL2 및 XYL3 과잉-발현(over-express)을 위해 유전자 조작된 S. 세레비시아 스트레인을 나타낸 것이다(XYL1 대 GRE3).
도 53 은 40 g/L 의 자일로스에서 성장한 유전자 조작된 S. 세레비시아 에 의한 자일로스 발현에서 과잉-발현된(over-expressing) XYL1 대 GRE3 의 효과를 나타낸 것이다.
도 54 은 80 g/L 의 자일로스에서 성장한 유전자 조작된 S. 세레비시아 에 의한 자일로스 발현에서 과잉-발현된(over-expressing) XYL1 대 GRE3 의 효과를 나타낸 것이다.
도 55 는 상이한 야생형 LAD 효소의 열 특성(thermal property) 및 pH-의존적인 특성(pH-dependent properties)을 나타낸 것이다 : LAD(■), tlLAD(◆) 및 pcLAD(●). (A) 온도-의존적인 촉매 활성도(Temperature-dependent catalytic activities), (B) 오버타임(over time) 동안의 50 ℃ 에서의 열의 불활성화(Thermal inactivation) 및 (C) pH-의존적인 촉매 활성도. 에러바(Error bars)는 평균의 표준 오차(standard error)를 나타낸다(n = 3).
도 56 은 N. 크라사 및 P. 스티피티스(psXDH)로부터의 XDH 의 정렬(alignment)을 나타낸 것이다.
도 57 은 N. 크라사 LAD 및 XDH 의 pH 비율 프로파일(pH rate profiles)의 비교를 나타낸 것이다. LAD 의 특성으로부터의 획득한 데이터는 일반적인 완충용액 MES/트리스/글리신에서 수행하였고, 낮은 pH 값에 대해 일반적인 완충용액 아세트산/MES/트리스에서 수행한 ncXDH[폐쇄된 삼각형(closed triangles)] 및 ncLAD[폐쇄된 원형(closed circles)] 에 대한 데이터는 중복되었다.
도 58 은 박테리오드 스테코리스(Bacteroids stercoris , BtXI), 비피도박테리움 롱검(Bifidobacterium longum , BfXI) 및 코돈-최적화된 BtXI(codon-optimized BtXI)에 대한 BtXIO 로부터의 자일로스 이성질화효소(xylose isomerase enzyme)를 갖는 형질전환된(transformed) S. 세레비시아 스트레인 L2612 에 의한 에탄올 생산을 나타낸 것이다. XI 유전자는 pRs424TEF 벡터 내로 클론되었다(clone).
도 59 는 벡터 pRS403TEF 에 의해 이의 게놈(genome) 내로 통합된(integrated) BtXI 를 갖는, S. 세레비시아 스트레인 D452-2 에 의한 자일로스 소비 및 에탄올 생산을 나타낸 것이다. 플라스미드로부터 BtXI 가 발현된 S. 세레비시아 스트레인 L2612 에 의한 자일로스-발효와 비교하였다.
도 60 은 통합된 BtXI 및 발현된 XYL2 또는 XYL3 또는 XYL2 및 XYL3 을 함유하는 S. 세레비시아 스트레인에 의한 자일로스 발효를 나타낸 것이다.
도 61 은 자일로스의 효율적인 대사작용을 위해 오탄당 인산경로(pentose phosphate pathway)에서 포함된 GND1 과 같은 과잉-발현된 효소를 유전자 조작한 S. 세레비시아 에서 XYL3 발현의 필요성을 나타낸 것이다.
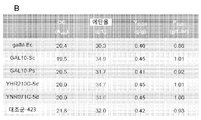
도 62 는 발효 파라미터(fermentation parameters)에서 대장균, S. 세레비시아 및 P. 스티피티스에서 NCU09705 상동성의 과잉-발현의 효과를 나타낸 것이다. (A) 셀로비오스 소비, 성장 및 에탄올 생산 ; 및 (B) 에탄올 수득률 및 생산성에서 galM, GALIO-Sc, GALIO-Ps, YHR210C 및 YNR071C 의 과잉-발현.
도 63 은 곰팡이(N. 크라사)로부터의 셀로덱스트린 동화 경로(assimilation pathway) 및 S. 세레비시아 내의 자일로스-발효 효모 P. 스티피티스로부터의 변형된 자일로스 대사경로의 통합을 통해서 글루코스 리프레션(repression)없이 셀로비오스 및 자일로스의 동시적인 공동-발효(co-fermentation)를 가능하게 하는 실험 설계를 나타낸 것이다. (A) 두 개의 대사작용이 가능하지 않는 당(non-metabolizable sugars)(셀로비오스 및 자일로스)을 발효시킬 수 있는 효모 스트레인을 유전자 조작하기 위한 스트레인 개선 전략(strain improvement strategy). 셀로덱스트린 동화 경로는 N. 크라사로부터의 셀로덱스트린 운반체(NCU00801) 및 세포 내의 β-글루코시다아제(NCU00130)으로 이루어져 있다. 변형된 자일로스 대사 경로는 자일로스 환원효소 동질효소(xylose reductase isozymes)(야생형 XR 및 돌연변이체 XR R.276H), 자일리톨 탈수소효소(XYL2) 및 자일루로키나아제(xylulokinase)(XKS1)를 사용하였다. (B) 유전자 조작된 S. 세레비시아에 의한 글루코스 및 자일로스를 함유하는 당 혼합물의 발효 프로파일(Fermentation profile)은 이러한 연구에서 진전되었다(develop). 글루코스가 고갈된 후에만 자일로스 발효가 시작되기 때문에 글루코스 발효는 자일로스 발효를 억제한다. (C) 유전자 조작된 S. 세레비시아에 의한 셀로비오스 및 자일로스를 함유하는 당 혼합물의 발효 프로파일은 이러한 실험에서 진전되었다. 탄소원(carbon source)이 다른 물질의 소비를 억제하지 않음에 따라 셀로비오스 및 자일로스는 동시에 사용되었다.
도 64 는 플라스미드 구성(plasmid construction)에 대한 도식(scheme)을 타나낸 것이다. pRS425 셔틀벡터(shuttle vector)는 DNA 조립 방법(DNA assembler method)(Shao 등의, 2009)를 사용한 셀로비오스 운반체 및 β-글루코시다아제 유전자를 조립한 다음에 선 모양으로 만들었다(linearized).
도 65 는 시간의 함수(function)으로서의 S. 세레비시아 스트레인 (A) SL01, (B) SL04, (C) SL02, (D) SL05, (E) SL03, (F) SL06 및 (G) SL00 에 의한 4 % 셀로비오스 및 5 % D-자일로스의 공동-발효 동안의 셀로비오스(■), 글루코스(●), D-자일로스(▲), 에탄올(▼), 및 바이오매스(□)의 농도의 변화를 나타낸 것이다.
도 66 은 시간의 함수로서 도표로 나타낸, 진탕-플라스크(a,b) 또는 바이오리액터(bioreactors)(c, d)에서 셀로비오스-자일로스 혼합물에서 성장한 S. 세레비시아 스트레인 SL01(a, b) 및 SL00(b, d)에서의 셀로비오스(■), 글루코스(●), D-자일로스(▲), 에탄올(▼), 및 바이오매스(□)의 농도의 변화를 나타낸 것이다.
도 67 은 시간의 함수로서 도표로 나타낸, 바이오리액터에서 5 g/L 글루코스-40 g/L 셀로비오스-50 g/L 자일로스 혼합물(a, b) 또는 10 g/L 글루코스-40 g/L 셀로비오스-50 g/L 자일로스 혼합물(c, d)를 함유하는 배지에서 성장한 S. 세레비사이 스트레인 SL01(a, c) 및 SL00(b, d)에서의 셀로비오스(■), 글루코스(●), D-자일로스(▲), 에탄올(▼), 및 바이오매스(□)의 농도의 변화를 나타낸 것이다.
도 68 은 (A) NCU00801, (B) NCU00809 및 (C) NCU08114 가 발현된 β-글루코시다아제(NCU00130)-함유 S. 세레비시아 스트레인에 의한 셀로비오스 활용의 비교를 나타낸 것이다. 부호 : 셀로비오스(■), 에탄올(◆) 및 OD600(●).
도 69 는 두 가지의 당의 다양한 농도를 함유하는 혼합물에서 성장한 S. 세레비시아 스트레인 DA24-16BT3 에 의한 셀로비오스 및 자일로스의 공동-발효를 나타낸 것이다 : (A) 20 g/L(각각)의 셀로비오스 및 자일로스, (B) 30 g/L(각각)의 셀로비오스 및 자일로스, 및 (C) 40 g/L(각각)의 셀로비오스 및 자일로스. 부호 : 셀로비오스(▲), 자일로스(■), 에탄올(◆) 및 OD600(●).
도 70 은 S. 세레비시아 스트레인 DA24-16BT3 에 의한 셀로비오스 및 자일로스의 공동-발효의 상승효과(synergistic effect)를 나타낸 것이다. 부호 : 셀로비오스(▲), 자일로스(■), 에탄올(◆) 및 OD600(●) : (A) 40 g/L 셀로비오스, (B) 40 g/L(각각)의 셀로비오스 및 자일로스, 및 (C) 40 g/L 자일로스.
도 71 은 S. 세레비시아 스트레인 DA24-16BT3 및 야생형 P. 스티피티스 스트레인에 의한 글루코스, 셀로비오스 및 자일로스의 공동-발효를 나타낸 것이다. 부호 : 셀로비오스(▲), 자일로스(■), 에탄올(◆), OD600(●) 및 글루코스(▼). (A) DA24-16BT3 및 (B) P. 스티피티스.
도 72 는 S. 세레비시아 스트레인 DA24-16BT3 에 의한 셀로비오스 및 자일로스의 c-발효 동안 셀로트리오스 및 셀로테트라오스 축적(accumulation)을 시사하는, 각각의 시점으로부터의 HPLC 크로마토그램(HPLC chromatograms)을 나타낸 것이다.
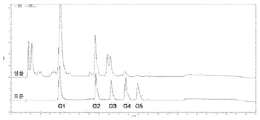
도 73 은 셀로비오스 및 자일로스의 공동-발효 동안의 S. 세레비시아 스트레인 DA24-16BT3 에 의한 22 시간 발효 후의 발효 배지에서 셀로덱스트린 축적을 보여주는 HPAEC 분석을 나타낸 것이다. [Gl : 클루코스, G2 : 셀로비오스, G3 : 셀로트리오스, G4 : 셀로테트라오스, 및 G5 : 셀로펜타오스(cellopentaose)].
도 74 는 40 g/L(각각)의 셀로비오스 및 자일로스의 공동-발효 동안에, 다중-복제 플라스미드(multi-copy plasmid)에서의 (A) NCU00801 의 통합된 복제(integrated copy) 및 (B) NCU00801 이 발현된 S. 세레비시아 형질전화체(S. cerevisiae transformants)에 의한 당 이용의 비교를 나타낸 것이다. 부호 : 셀로비오스(▲), 자일로스(■), 에탄올(◆), 및 OD600(●).
도 75 는 (A) 연구에서 사용된 두 가지 상이한 S. 세레비시아 스트레인 : DA24-16 및 D452BT, 두 가지 상이한 효모 스트레인의 배양(cultivation)에 의한 에탄올 생산을 나타낸 것이다. 자일로스 분자는 오각형으로 나타내었고 셀로비오스 분자는 두 개의 육각형으로서 나타내었다 ; 및 (B) 자일로스-발효 스트레인 및 셀로비오스-발효 스트레인의 혼합된 배양물(Mixed cultures).
도 76 은 N. 크라사에서 354 자일란-유도 유전자(354 xylan-induced genes)의 목록을 나타낸 것이다.
도 77 은 다양한 N. 크라사 녹-아웃 스트레인(knock-out strains)에 대한 분비된 단백질 수준(secreted protein levels), 감소된 당(reducing sugar) 및 아조-자일라나아제 활성도(azo-xylanase activity)를 나타낸 것이다. 분비된 단백질 수준은 모든 스트레인에 대해서 상대적인 상수(relatively constant)이다.
도 78 은 (A) 야생형, △NCU05137 및 △NCU05137/△NCU05137-GFP 네우로스포라 스트레인에 대한 총 분비된 단백질 및 CMC-활성도, 및 (B) 세 가지 상이한 스트레인의 배양물(cultures)의 상청액(supernatant)에서의 총 단백질의 쿠마시 염색(Coomassie strain)을 나타낸 것이다.
도 79 는 분생자(conidia)에서 NCU05137-GFP 의 위치(localization)를 나타낸 것이다.
도 80 은 균사 팁(hypha tip)에서 NCU05137-GFP 의 위치를 나타낸 것이다.
도 2 는 NCU05137 의 계통분석(phylogenetic analysis)을 나타낸다. 엔. 크라사 NCU05137 의 예측된 오솔로그(predicted orthologs)는 BLAST 에 의한 중요한 유사성을 나타내는 아미노산 서열을 기초로 한 NCBI 및 JGI 로부터 검색하였다. 모든 확인된 실모양의 진균의 오솔로그(filamentous fungal orthologs)를 나타낸다 ; NCBI E 수치는 9e-175 인 B. 푸켈리아나(B. fuckeliana)를 제외한 0.0 이다. NCU05137 의 동족체(Homologs)는 수많은 박테리아에서 또한 확인된다(E 값 ~e-30). 폴라로모나스 나프탈렌이보란스(Polaromonas naphthalenivorans)(베타-프로테오박테리움, beta-proteobacterium)로부터의 YP 981875 는 외집단(outgroup)으로서 사용되었다. A. = 아스페르길루스(Aspergillus) ; N. = 네오사토리아(Neosartorya) ; P. 키로소게늄(P. chyrosogenum) = 페니실리움(Penicillium) ; S. = 스클레오티니아(Sclerotinia) ; B. = 보트리요티니아(Botryotinia) ; P. = 피레노포라(Pyrenophora) ; C. = 코칠레오볼루스(Cochliobolus) ; N. 하마토코카(N. haematococca) = 넥트리아(Nectria) ; P. 안세리나(P. anserina) = 포도스포라(Podospora) ; N. = 네우로스포라(Neurospora). 나무형(tree)은 MEGA3, NJ 에 의해 생성되었다. 아미노산 사이트 당 바(bar) = 0.2 치환(substitutions).
도 3 은 단일 탄소원(sole carbon source)으로서의 미스칸터스 ( Miscanthus ) 및 아비셀(Avicel)에서 성장하였을 때의 N. 크라사 FGSC2489 및 T. 레세이(T. reesei) QM9414 엔도글루카나제 활성도(endoglucanase activity)의 분석을 나타낸 것이다. N. 크라사 WT 계통(strain) FGSC2489 및 T. 레세이(T. reesei) QM9414 의 배양 여과물에서 엔도글루카나제 활성도. N. 크라사는 25 ℃ 에서 단일 탄소원으로서의 2 % 의 아비셀 또는 억세 파우더를 함유하는 Vogel's 최소배지에서 성장하였다. T. 레세이 계통은 25 ℃ 에서 단일 탄소원으로서의 1 % 의 아비셀 또는 억세 파우더를 갖는 MA 배지에서 접종하였다(inoculate). 둘 다의 계통을 동일한 양의 분생자(conidia)[lOO mL 배양물(culture)에 1 x 106 /mL]로 접종하였다. 배양물에서 상이한 시점에서의 엔도글루카나제 활성도를, 제조자의 지시(manufacturer's instructions)(아일랜드에 위치한 Megazyme)에 따라 기질(substrate)로서 Azo-CM-셀룰로스를 사용하여 pH 4.5 에서 측정하였다.
도 4 는 미스칸터스 및 아비셀에서 성장한 N. 크라사의 전사 프로파일링(transcriptional profiling)을 나타낸 것이다. (A) 미스칸터스 배양에서 발현의 차이점으로 나타내는 769 유전자의 계층적 군집 분석(Hierarchical clustering analysis). 어두운 셰이딩(Dark shading)은 상대적으로 보다 높은 발현(expression)을 나타내고, 밝은 셰이딩(light shading)은 상대적으로 낮은 발현을 나타낸다. 라인 1(Lane 1) : 16 시간 동안의 Vogel's 최소 배지 N. 크라사 배양물(Vogel 1956)의 발현 프로파일(Expression profile). 라인 2 : 16 시간 동안의 단일 탄소원으로서의 미스칸터스에서 성장한 배양물의 발현 프로파일. 라인 3, 4, 5 : 40 시간, 5 일 및 10 일 동안의 미스칸터스에서 성장한 배양물로부터의 발현 프로파일. 세 가지 군집(clusters)을 C1, C2 및 C3 으로 나타내었다. 대부분의 셀룰라아제 및 헤미셀룰라아제 유전자의 증가된 발현 수준을 나타낸 군집은 상자 모양을 나타내었다(boxed)(C3 군집). (B) N. 크라사 미스칸터스 대 아비셀 성장한 배양물 사이의 발현 프로파일에서 공통부분(overlap)의 분석. 단백질의 분석 및 공통부분은 이중 질량 분석기(tandem mass spectrometry)(Bottom)로 미스칸터스 및 아비셀에서 성장한 N. 크라사의 배양 여과물에서 측정하였다. (C) 미스칸터스 배양물에서 상대적인 발현 수준에서 증가됨을 나타낸 231 유전자의 기능범주(Functional category, FunCat) 다양한 분석(enrichment analysis)(Ruepp 2004). 분류되지 않은 그룹(the unclassified group)을 포함한 중요한 풍부함(enrichment)(p<0.001)을 나타낸 기능범주를 나타낸다.
도 5 는 최소배지(MM)에서 성장하는 동안 및 16 시간(16 hr), 40 시간(40 hr), 5 일(5 days) 및 10 일(10 days) 동안 미스칸터스(Miscanthus)에서 성장하는 동안의 셀룰라아제(A) 및 헤미셀룰라아제(B)를 코딩하는 N. 크라사 유전자의 상대적인 발현 수준을 나타낸다.
도 6 은 MS 에 의해 확인된 분비된 단백질을 코딩하는 유전자의 삭제를 포함하는 스트레인(strains)으로부터의 배양 상청액(culture supernatants)의 단백질 프로파일(protein profile) 및 효소 활성도(enzymatic activity)를 나타낸 것이다. (A) 7 일 동안 아비셀에서 성장한 야생형(wild type)과 비교한 16 결실 스트레인(deletion strains)의 배양 여과물(culture filtrates)에 나타난 단백질의 SDS-PAGE. 삭제 스트레인(strain)은 미스칸터스 및 아비셀 배양 여과물 둘 다에서의 질량 분석법(mass spectrometry)에 의한 단백질의 식별(identification)을 기반으로 선택되었다. 스트레인은 유전자 NCU 번호를 기초로 정리하였고, 야생형 스트레인(wild-type strain)은 FGSC 2489 이다. 삭제된 유전자에 해당하는 누락된 단백질 밴드(Missing protein bands)는 상자모양으로 나타났다(boxed). (B) 총 분비된 단백질, 아조-CMCase(azo-CMCase) 및 β-글루코시다아제 활성도 검정(실시예 5 참고)을 (A)로부터의 동일한 샘플을 사용하여 16 삭제 스트레인 및 야생형 부모 스트레인(wild-type parental strain)(FGSC 2489)에서 실행하였다. 활성도 및 단백질 농도를 야생형 농도과 비교하여 정규화시키고, 3 번의 생물학적인 측정(triplicate biological measurements)의 평균을 나타내었다. (C) (A) 에서의 동일한 샘플을 사용한 16 삭제 스트레인로부터의 배양 여과물의 셀룰라아제 활성도. 아비셀라아제 활성도(Avicelase activity)를 측정하기 위해 배양 여과물(Culture filtrates) 10 배 희석하였고 5 mg/mL 아비셀과 혼합하였다(실시예 5 참고). 40 ℃ 에서 배양하여 8 시간 후에 글루코스(흑색) 및 셀로비오스(cellobiose)(백색)를 측정하였다.
도 7 은 단일 탄소원으로서 아비셀에서 성장한 배양물로부터의 돌연변이체 분석을 기반으로 한 N. 크라사 분비된 단백질의 정체(identity)를 나타낸 것이다. 25 ℃ 에서 7 일 동안 100 mL 의 진탕 플라스크(shake flasks)에서 2 % 아비셀에서 성장한 WT N. 크라사(FGSC 2489)로부터의 분비된 단백질의 SDS-PAGE. 15 μL 의 농축시키지 않는 배양 여과물을 크라이테리언 4-15 % 26-웰 겔 내에 로딩하였다(loaded). 내셔널 진단(National Diagnostics)으로부터는 포토 블루 세이프(Proto Blue Safe)(Coomassie)를 겔을 착색시키는데(stain) 사용하였다. 단백질 밴드를, 결실 스트레인(deletion strains)에서 분비된 단백질의 분석을 기반으로 한 도 6A 에 나타낸 바와 같은 이러한 연구에서 확인하였다.
도 8 은 △NCU04952 및 △ANCU05137 에서 cbh-1(NCU07340) 및 gh6-2 (NCU09680; CBHII)의 발현 및 분비된 단백질 프로파일을 나타낸 것이다. (A) WT, △NCU04952, 및 △NCU05137 에서 총 분비된 단백질의 SDS-PAGE. 배양물을 분생자(conidia)로부터의 아비셀에서 배양물을 성장시키고 30 시간, 2 일(48 시간) 및 3 일(72 시간)에 채취하였다(harvested)(실시예 5 참고). 라인 1-3 은 각각의 WT, △NCU04952 및 △NCU05137 스트레인(strains)로부터의 아비셀에서 30 시간 성장 후의 20x 농축시킨 배양 여과물이다. 라인 4-6 은 각각의 WT, △NCU04952 및 △NCU05137 스트레인로부터의 아비셀에서 2 일 성장 후의 농축시키지 않은 배양 여과물이다. 라인 7-9 는 각각의 WT, △NCU04952 및 △NCU05137 스트레인으로부터의 3 일후의 농축시키지 않은 배양 여과물이다. (B) 아비셀에서 성장하는 동안의 WT, △NCU04952 및 △NCU05137 스트레인에서 cbh -1(NCU07340; CBHI) 및 gh6-2(NCU09680; CBHII)의 RT-PCR. WT 및 삭제 스트레인은 분생자로부터의 아비셀에서 성장하였고, 48 시간 및 72 시간에 채취하였다(실시예 5 참고). 단일 탄소원으로서의 수크로스를 갖는 최소배지(MM) 배양물(Vogel 1956)을 16 시간 동안 성장시켰다[유사한 발달 시점(developmental time point)]. cbh -1 및 gh6-2 의 폴드 인덕션(fold induction)은, 모든 샘플의 대조군으로서 사용된 액틴 유전자 발현(actin gene expression)을 갖는 MM 조건 하에서 이러한 유전자의 발현과 관련되어 있다.
도 9 는 N. 크라사에서 식물 세포 벽 디컨스트럭션(deconstruction)의 모델을 나타낸 것이다. 도입(induction) : 낮은 수준에서 발현된 세포외 효소(Extracellular enzymes)는 대부분 분비된 식물 세포 벽 분해효소(plant cell wall degrading enzymes)를 코딩하는 유전자의 발현 수준을 극적으로 증가시키기 위해 단일 N. 크라사의 2 차 대사물질(secondary metabolites)를 생성한다. 이용(utilization) : 세포 벽 분해산물(cell wall degradation products)의 전좌(translocation)에 특이적인 세포외 효소 및 운반체(transporters)는 N. 크라사가 성장에 대한 식물 세포 물질을 활용할 수 있게 한다. 몇몇의 세포외 단백질(NCU05137, NCU05057, 및 NCU04952)은 활용 단계에서 셀룰라아제(cellulases) 및 헤미셀룰라아제(hemicellulase )의 유전자 발현을 조절하는 대사물질(metabolites)을 생성할 수 있다 ; 이중의 육각형(double hexagon)(셀로비오스), 이중의 오각형(double pentagon)(자이로비오스, xylobiose), 육각형(글루코스) 및 오각형(자일로스). 그려진 식물 세포 벽-분해 효소는 CBH(I), CBH(II), EG2, EG1, EG6 및 자일라나아제(xylanase)를 포함한다. 추가적인 셀룰로이스 분해효소(cellulolytic enzymes)를 나타내지 않는다. 화살표의 두께는 반응의 상대적인 강도를 나타낸다.
도 10 은 S. 서머파일 단백질 서열(S. thermophile protein sequences)의 데이터베이스에 대한 N. 크라사 추정상의 운반체(putative transporters)의 서열 또는 N. 크라사 단백질 서열의 데이터베이스에 대한 S. 호열성 추정상의 운반체의 서열을 BLAST 에서 검색한 결과를 나타낸 것이다.
도 11 은 NCU08114 가 결핍된 N. 크라사 스트레인의 성장 표현형(growth phenotype)을 나타낸 것이다. (A) 탄소원으로서 결정질 셀룰로스(crystalline cellulose)를 갖는 성장의 3 일 후의 WT(왼쪽) 및 △NCU08114(오른쪽) N. 크라사 스트레인의 진탕 플라스크(Shaker flasks). (B) 각각의, 16 시간 또는 28 시간 동안, 탄소원으로서 수크로스 또는 결정질 셀룰로스을 이용하여 성장한 N. 크라사 배양물로부터의 평균 알라마 블루ⓒ 형광(Mean Alamar Blueⓒ fluorescence). 형광을 100 % 로 WT 에 맞추어 정규화시켰다(normalize). 에러 바(Error bars)를 세 번의 생물학적 반복(biological replicates)으로부터의 측정(measurements) 사이의 표준 편차(standard deviation)이다. NCU00801이 결핍된 N. 크라사는, 단당류 운반체에 의해 순차적으로 계속되는 셀로덱스트린에서 글루코스로 가수분해하는(Scarborough 1973) N. 크라사 분비된 β-글루코시다아제의 명백한 표현형을 갖지 않는다(Tian 등의, 2009). 이러한 소비(consumption)의 우회 루트(alternate route)는 이러한 삭제 라인(deletion lines)에서 셀로덱스트린 운반체의 결함의 과소평가(underestimate)로 이어진다.
도 12 는 (A) NCU00130 와 함께 NCU00801, NCU05853 또는 NCU08114 가 발현된 S. 세레비시아 스트레인(S. cerevisiae strains)에 대한 셀로비오스(cellobiose)의 소비 ; (B) NCU00130 와 함께 NCU00801, NCU05853 또는 NCU08114 가 발현된 S. 세레비시아 스트레인에 대한 셀로트리오스(cellotriose)의 소비 ; (C) NCU00130 와 함께 NCU00801, NCU05853 또는 NCU08114 가 발현된 S. 세레비시아 스트레인에 대한 셀로테트라오스(cellotetraose)의 소비 ; 및 (D) NCU00130 와 함께 NCU00801, NCU05853 또는 NCU08114 가 발현된 S. 세레비시아 스트레인에 대한 셀로헥사오스(cellohexaose)의 소비를 나타낸 것이다.
도 13 은 NCU008114 또는 NCU00801 이 결핍된 N. 크라사 스트레인에 의한 셀로덱스크린의 소비를 나타낸 것이다. 나타낸 N. 크라사 스트레인을 15 분 동안 90 μL 의 각각의 당으로 배양하였다. 바(Bars)는 두 개의 독립적인 실험으로부터의 배양 후에 상청액에 남아있는 당의 평균 농도를 나타낸 것이다. 에러 바는 이들 실험 사이의 표준 편차이다.
도 14 는 NCU00801/cbt1 이 발현된 S. 세레비시아 스트레인(S. cerevisiae strain)에 의한 셀로비오스 수송을 나타낸 것이다. CBT 1 이 있을 때(○) 또는 없을 때(●)에 의한 셀로비오스 수송을 나타낸 것이다. 스트레인 둘 다는 세포 내의 β-글루코시다아제, NCU00130 가 발현되었다. 셀로비오스의 초기 농도는 50 μM 이다. 모든 값은, 이러한 측정 사이의 표준 편차를 나타낸 에러 바와 함께 두 측정 사이의 평균이다.
도 15 는 CBTl 및 CBT2 에 융합시킨(fused) GFP 의 위치(localization) 및 정량(quantification)을 나타낸 것이다. (A) 이들의 C-말단에 GFP 에 융합시킨 cbtl(왼쪽) 또는 cbt2(오른쪽)에 발현된 S. 세레비시아 스트레인의 이미지. (B) 셀로비오스 운반체가 없는 또는 이들의 C-말단에 GFP 에 융합시킨 cbtl 또는 cbt2 가 발현된 효모 스트레인의 GFP 형광(fluorescence). 값은 세 번의 생물학적 반복으로부터의 평균이고, 에러 바는 이들의 반복 사이의 표준 편차를 나타낸다.
도 16 은 S. 세레비시아에서 발현된 N. 크라사 수송 시스템에 의한 셀로덱스트린 수송을 나타낸 것이다. (A) 유전자 NCU00801(cbtl 으로 명명, ○), NCU08114 (cbt2 로 명명, ▼)가 발현되거나, 또는 운반체가 없는 경우(●)의 효모 스트레인의 셀로비오스-매개 성장(Cellobiose-mediated growth). 모든 스트레인은 또한 세포 내의 β-글루코시다아제, NCU00130 를 발현하였다. 대표적인 실험을 나타내었다. 세 가지의 독립적인 실험으로부터의 성장비율을 하기와 같다 : cbtl, 0.0341 ± 0.0010 hr-1; cbt2, 0.0131 ± 0.0008 hr-1 ; 운반체가 없는 경우, 0.0026 + 0.0001 hr-1. (B) 셀로트리오스 및 셀로테트라오스에서 효모 스트레인의 성장. 범례(legend)에 열거된 운반체 뿐만 아니라 세포내의 β-글루코시다아제, NCU00130 가 발현된 스트레인은 단일 탄소원으로서 제공된 0.5% (w/v)의 셀로트리오스(G3) 또는 셀로테트라오스(G4)와 함께 성장시켰다. 대표적인 실험을 나타내었다. 세 가지 독립적인 실험으로부터의 성장율(Growth rates)은 하기와 같다 : cbtl 셀로트리오스, 0.0332 + 0.0004 hr-1; cbtl 셀로테트라오스 0.0263 + 0.0020 hr-1 ; 운반체가 없는 셀로트리오스, 0.0043 + 0.0015 hr-1; cbt2 셀로트리오스, 0.0178 + 0.0005 hr-1; cbt2 셀로테트라오스 0.0041 + 0.0003 hr-1; 운반체가 없는 셀로테트라오스, 0.0031 + 0.0008 hr-1. (C) 글루코스는 정제된 NCU00130 에 의해 셀로비오스(G2), 셀로트리오스(G3) 및 셀로테트라오스(G4) 가수분해로부터 생산되었다. 세 가지의 독립적인 측정의 평균 및 표준 편차를 나타내었다. 효소(2 nmol)가 없는 배양에서의 잔류 글루코스는 나타낸 값으로부터 뺐다.
도 17 은 글루코스에서 운반체가 없거나(●) 또는 cbt1(○), cbt2(▼)가 발현된 S. 세레비시아 스트레인의 성장을 나타낸 것이다. 모든 스트레인은 β-글루코시다아제, NCU00130 이 발현되었다. 대표적인 실험을 나타내었다.
도 18 은 250 mL 의 플라스크에서 S. 세레비시아 스트레인의 셀로비오스-매개 성장을 나타낸 것이다. 값은 β-글루코시다아제, NCU00130, cbtl 또는 cbt2 가 발현된 효모 스트레인, 또는 어떠한 운반체도 결핍된 스트레인 발현 NCU00130 의 두 번의 복제 배양물(two replicate cultures) 사이의 평균 OD 를 나타낸 것이다. 에러 바는 복제 사이의 표준 편차를 나타낸다.
도 19 는 CBT1 및 CBT2 에 의한 셀로비오스 수송의 반응속도(kinetics)를 나타낸 것이다. 셀로비오스 수송의 비율은 cbtl 또는 cbt2 가 발현된 효모 스트레인에 의한 셀로비오스 농도의 작용으로서 측정하였다. 수송 비율(transport rate)은 풍부한 운반체(transporter abundance)에 대해서 정규화시켰다(normalize).
도 20 은, 셀로비오스, 셀로트리오스 또는 셀로테트라오스에서 성장한 x-축에 나타낸 네우라스포라 유전자(Neurospora genes)의 조합을 발현된 S. 세레비시아의 능력을 나타낸 것이다.
도 21 은 cbtl 또는 cbt2 를 운반하는 스트레인에서 셀로비오스 수송을 위한 셀로덱스트린의 경쟁(competition)을 나타낸 것이다. 각각의 분류되지 않은 당(unlabeled sugar)의 5-폴드 초과량(5-fold excess)은 [3H]-셀로비오스 수송의 검정 동안에 포함되었다. CBT1 또는 CBT2 의 기질(Substrate)은 결합을 위한 경쟁에 의해 [3H]-셀로비오스 수송 비율을 감소시켰다. 바는 세 번의 반복으로부터의 평균을 나타낸다. 에러 바는 이러한 반복 사이의 표준 편차를 나타낸다. 값은 100 까지의 경쟁 당 없이 [3H]-셀로비오스 수송 비율에 맞추어 정규화시켰다.
도 22 는 정제된 NCU00130 의 SDS-PAGE 겔을 나타낸 것이다. 라인 1, kDa 에서의 단백질 분자량 기준. 라인 2, 니켈-NTA 레진(nickel-NTA resin) 상에서 여과한 후의 NCU00130. kDa 의 분자량은 왼쪽에 나타내었다.
도 23 은 셀로비오스 운반체 NCU08114 및 NCU00801 의 최고 가능성을 갖는 계통발생론적 분석(maximum likelihood phylogenetic analysis)을 나타낸 것이다. S. 세레비시아 HXT1 및 K. 락티스 LACP 를 예외로 하는, 나타낸 단백질을 코딩하는 모든 유전자는, 균류(fungus)가 식물 세포 벽 물질 또는 셀로비오스와 접촉하여 쓰이기 시작할 때, 발현 수준을 증가시키기 위한 것으로 알려졌다(Tian 등의, 2009; Noguchi 등의, 2009; Wymelenberg 등의, 2010; Martin 등의, 2010). S. 세레비시아 HTX1, 낮은 친화도(low affinity)의 글루코스 운반체(Reifenberger 등의, 1997)는 외집단(outgroup)으로서 사용되었다.
도 24 는 셀로비오스의 발효(fermentation), N. 크라사로부터의 셀로비오스 수송 시스템이 발현된 S. 세레비시아에 의한 동시의 당화(saccharification) 및 셀룰로스의 발효를 나타낸 것이다. (A) 에탄올에 대한 셀로비오스 발효. 에탄올은 CBTl 을 갖거나(●) 또는 CBTl 을 갖지 않은(○) 효모 스트레인에 의해 생산되었다. CBTl 을 갖거나(▼) 또는 CBTl 을 갖지 않은(△) 효모 스트레인을 사용한 발효 반응 동안에 셀로비오스 농도. (B) CBTl 을 갖거나, 갖지 않은 효모 스트레인을 사용한 SSF. CBTl 을 갖는 스트레인의 존재 하에서 셀로비오스(●) 및 글루코스(▼)의 농도, 및 CBTl 이 결핍된 스트레인의 존재 하에서 셀로비오스(○) 및 글루코스(△)의 농도. 참고로 0.1 mg/mL 셀로비오스 = 292 μM. (C) CBTl 을 갖는(●) 또는 CBTl 을 갖지 않는(○) 스트레인을 사용한 SSF 동안 생산된 에탄올. 모든 패널(panels)에서, 값은 세 번의 생물학적인 반복의 평균이다. 에러 바는 이러한 반복 사이의 표준 편차이다. 모든 스트레인은 세포 내의 β-글루코시다아제, NCU00130 을 발현시켰다.
도 25 는 효모에 의한 셀롤로스의 동시의 당화(simultaneous saccharification) 및 발효 동안에 곰팡이(filamentous fungi)로부터의 셀로덱스트린 수송 경로(transport pathways)의 사용을 나타낸 것이다. 셀로덱스트린(Cdes) 수송 경로(흑색)은 셀로덱스트린 운반체(CBT) 및 세포 내의 β-글루코시다아제(βG)를 포함한다. 표준 효모(standard yeast)에 나타낸 당 이화 경로(sugar catabolism pathway)는 육탄당 운반체(hexose transporters, HXT)를 포함한다. SSF 에서 셀룰라아제(GH) 및 세포외 β-글루코시다아제(βG) 둘 다는 사용될 수 있다.
도 26 은 기능적으로 중요한 NCU00801 및 NCU0811 에서의 잔여물(residues)을 나타낸 것이다. (A) cbtl/NCU00801 의 Ala-스캔(Ala-scan). (B) cbtl NCU00801 의 폴리펩티드 서열[표시된 중요한 잔기(important residues marked)]. (C) cbt2/NCU08l14 의 폴리펩티드 서열(표시된 중요한 잔기).
도 27 은 P. 스티피티스(P. stipitis)로부터의 셀로비오스 운반체가 발현된 S. 세레비시아 스트레인의 비교를 나타낸 것이다. (A) β-글루코시다아제가 발현된 S. 세레비시아 스트레인 및 셀로비오스 운반체 NCU00801, NCU08114 및 NCU05853 의 오솔로그(orthologs)의 세포 성장. (B) P. 스티피티스로부터의 셀로비오스 운반체의 비교 : β-글루코시다아제 및 셀로비오스 운반체가 발현된 S. 세레비시아 스트레인의 세포 성장. (C) P. 스티피티스로부터의 셀로비오스 운반체의 비교 : β-글루코시다아제 및 셀로비오스 운반체가 발현된 S. 세레비시아 스트레인에 의한 자일로스 소비 및 에탄올 생산.
도 28 은 셀로비오스 운반체 오솔로그의 배열을 나타낸 것이다. (A) 테스트된 조건 하에서 운반체 기능을 갖는 것으로 나타나지 않은 하나를 포함하는 셀로비오스 운반체 오솔로그의 배열. (B) 운반 기능을 갖는 셀로비오스 운반체 오솔로그의 배열. (C) NCU00801 및 NCU08114 의 배열.
도 29 는 NCU00801 및 NCU08114 의 상동성 모델(homology models)에서 표시된 기능적으로 중요한 모티프(motifs)를 나타낸 것이다. (A) NCU00801 의 상동성 모델에서 셀로비오스 운반체 모티프의 위치. 모티프는 흑색으로 강조되었다. 모티프 [LIVM]-Y-[FL]-x(13)-[YF]-D 를 페널(panels) a 및 b 에 나타내었다. 모티프 [YF]-x(2)-G-x(5)-[PVF]-x(6)-[DQ]를 페널 c 및 d 에 나타내었다. 모티프 G-R-[RK] 를 페널 e 및 f 에 나타내었다. 모티프 R-x(6)-[YF]-N 를 페널 g 및 h 에 나타내었다. 모티프 WR-[IVLA]-P-x(3)-Q 를 페널 i 및 j 에 나타내었다. 모티프 P-E-S-P-R-x-L-x(8)-A-x(3)-x(2)-Y-H 를 페널 k 및 l 에 나타내었다. 모티프 F-[GST]-Q-x-S-G-N-x-[LIV]를 페널 m 및 n 에 나타내었다. 모티프 L-x(3)-[YIV]-x(2)-E-x-L-x(4)-R-[GA]-K-G 를 페널 o 및 p 에 나타내었다. 참고 : 이러한 이미지에서, 트랜스멤브레인 헬릭스 6 및 7 을 연결하는 몇몇의 잔기는, 그들이 구멍을 가렸기 때문에(as they occlude the pore) 명확하게 제거되었다. (B) NCUQ8114 상동성 모델에서 셀로비오스 운반체 모티프의 위치. 모티프는 흑색으로 강조되었다. 모티프 [LIVM]-Y-[FL]-x(13)-[YF]-D 를 페널 a 및 b 에 나타내었다. 모티프 [YF]-x(2)-G-x(5)-[PVF]-x(6)-[DQ]를 페널 c 및 d 에 나타내었다. 모티프 G-R-[RK]를 페널 e 및 f 에 나타내었다. 모티프 R-x(6)-[YF]-N 을 페널 g 및 h 에 나타내었다. 모티프 WR-[IVLA]-P-x(3)-Q 를 페널 i 및 j 에 나타내었다. 모티프 P-E-S-P-R-x-L-x(8)-A-x(3)-x(2)-Y-H 를 페널 k 및 l 에 나타내었다. 모티프 F-[GST]-Q-x-S-G-N-x-[LIV]를 페널 m 및 n 에 나타내었다. 모티프 L-x(3)-[YIV]-x(2)-E-x-L-x(4)-R-[GA]-K-G 를 페널 o 및 p 에 나타내었다. 참고 : 이러한 이미지에서, 트랜스멤브레인 헬릭스 6 및 7 을 연결하는 몇몇의 잔기는, 그들이 구멍을 가렸기 때문에 명확하게 제거되었다.
도 30 은 플라스미드 발현의 구성에서 사용된 클로닝 과정(cloning process)을 나타낸 것이다 : (A) 추정상의 운반체(putative transporters) 및 (B) 운반체-GFP 융합 단백질(transporter-GFP fusion proteins).
도 31 은 글루코스-흡수 활성도(glucose-uptake activity)를 갖는 것으로 확인된 추정상의 운반체(putative transporters)의 오탄당 수송 활성도를 나타낸 것이다.
도 32 는 글루코스-흡수 활성도(glucose-uptake activity)를 갖지 않는 것으로 확인된 추정상의 운반체의 오탄당 수송 활성도를 나타낸 것이다.
도 33 는 NCU00821(AN25), STL12/XUT6(Xyp29) 및 XUTl(Xyp32)의 오탄당 흡수를 나타낸 것이다. A 는 자일로스 흡수를 나타내고 B 는 아라비노스 흡수를 나타낸 것이다.
도 34 는 STL12 XUT6 가 발현된 S. 세레비시아에 의한 l4C-표지된(labeled) 당 흡수를 나타낸 것이다(Xyp29).
도 35 는 GFP 형광에 의해 관찰된 바와 같은 S. 세레비시아 세포에서 발현된 운반체의 위치(localizations)를 나타낸 것이다. 첫 번째 줄의 왼쪽에서 오른쪽 : NCU00821-GFP 형광, NCU00821 핵(nuclei) ; 두 번째 줄의 왼쪽에서 오른쪽 : STL12 XUT6-GFP 형광, STL12/XUT6 핵.
도 36 은 (A) NCU00821(AN25), (B) STL12/XUT6(Xyp29) 및 (C) XUTl(Xyp32)으로 발현된 완충되지 않은 세포 현탁액에 말토오스의 첨가 상에서 pH 에서의 효과를 나타낸 것이다. 검정색 화살표는 말토오스를 첨가하였을 때의 시점(time points)을 나타낸 것이다.
도 37 은 NCU00821, STL12/XUT6 및 XUT1 의 공수송체 검정(syraporter assay)의 결과를 나타낸 것이다. (A) 자일로스에 대한 NCU00821. (B) 아라비노스에 대한 NCU00821. (C) 아라비노스에 대한 XUTl. (D) 자일로스에 대한 NCU00821. (E) 아라비노스에 대한 STL12 XUT6. (F) 자일로스에 대한 STLl2/XUT6. 검정색 화살표는 말토오스를 첨가하였을 때의 시점을 나타낸 것이다.
도 38 은 운반체의 과잉발현(overexpression)의 표현형 분석(phenotypic analyses)를 나타낸 것이다. (A) 는 OD 를 나타낸 것이고, (B) 는 자일로스 농도를 나타낸 것이고, (C) 는 0.5 % 자일로스-함유 배지에서의 자일로스 소비를 나타낸 것이다. (D) 는 OD 를 나타낸 것이고, (E) 는 자일로스 농도를 나타낸 것이고, (F) 는 0.5 % 자일로스-함유 배지에서의 자일로스 소비를 나타낸 것이다. (G) 는 다복제 플라스미드(multicopy plasmid), pRS424 에서 도입된 오탄당 운반체를 함유하는 S. 세레비시아의 성장 곡선을 나타낸 것이다.
도 39 는 이종 운반체(heterologous transporters)의 클로닝(cloning)에 사용된 플라스미드의 맵(maps)을 나타낸 것이다.
도 40 은 오탄당 운반체 오솔로그가 발현된 S. 세레비시아 스트레인에 의한 당-흡수 검정의 결과를 나타낸 것이다.
도 41 은 Clustal W (1.81)에 의한 오탄당 운반체 오솔로그의 서열 정렬(sequence alignments)을 나타낸 것이다. (A) 자일로스 운반체 오솔로그의 정렬. (B) 아라비노스 운반체의 정렬. (C) 자일로스 및 아라비노스 운반체의 정렬. 컨센서스 키(Consensus key) : * - 단일, 완전히 보존된 잔기(single, fully conserved residue) ; : - 강한 그룹(strong groups)의 보존(conservation) ; - 약한 그룹(weak groups)의 보존.
도 42 는 자일로스-이용 효소(xylose-utilizing enzymes)를 발현시키기 위한 제작한(engineered) 상이한 S. 세레비시아 스트레인을 나타낸 것이다.
도 43 은 자일로스 이용 경로 효소(xylose utilization pathway enzymes)를 함유하는 동일한 카세트(cassettes)가 발현된 상이한 백그라운드(backgrounds)의 세 가지 S. 세레비시아 스트레인의 자일로스 대사(xylose metabolism)(자일로스 소비 및 에탄올 생산 등과 같이 모니터 한 바와 같이)를 나타낸 것이다.
도 44 는 자일로스 이용 경로 효소를 함유하는 동일한 카세트를 발현하는 상이한 백그라운드의 세 가지 S. 세레비시아 스트레인의 자일로스-흡수 비율 및 대사물 수득률(metabolite yield)을 나타낸 것이다.
도 45 는 다양한 조건 하에서 S. 세레비시아 스트레인 DA24 에 의한 자일로스 발효(xylose fermentation)를 나타낸 것이다. (A) 진탕 플라스크에서 40 g/L 의 자일로스, (B) 진탕 플라스크에서 80 g/L 의 자일로스 및 (C) 바이오리액터(bioreactor)에서 80 g/L 의 자일로스. 부호 : 자일로스(■), 에탄올(◆) 및 OD600(●).
도 46 은 (A) S. 세레비시아 DA24 및 (B) P. 스티피티스 (P. stipitis ) 사이의 자일로스 소비 및 에탄올 생산의 비교를 나타낸 것이다. 부호 : 자일로스(■), 에탄올(◆) 및 OD600(●).
도 47 은 유전자 조작된(engineered) S. 세레비시아에서의 자일로스 대사 상에서 XYL2 과잉-발현 수준(over-expression levels)를 테스트하는데 사용된 실험 설계(experimental design)를 나타낸 것이다.
도 48 은 유전자 조작된(engineered) 자일로스-발효 S. 세레비시아 내로의 추가적인 XYL2 통합(integration)(즉, 증가된 XYL2 발현 수준)의 효과를 나타낸 것이다.
도 49 는 유전자 조작된 S. 세레비시아 에 의한 자일로스에서 XYL2 및 XYL3추가적인 동시의 과잉-발현(additional simultaneous over-expression)의 효과를 나타낸 것이다.
도 50 은 상이한 수준의 자일로스-발효 효소(xylose-fermenting enzymes)가 발현된 S. 세레비시아 스트레인을 나타낸 것이다.
도 51 은 유전자 조작된 S. 세레비시아 에 의한 차별된 XYL1 발현(differential XYL1 expression)의 효과를 나타낸 것이다.
도 52 는 상이한 환원효소(reductases)를 갖는 동일한 XYL2 및 XYL3 과잉-발현(over-express)을 위해 유전자 조작된 S. 세레비시아 스트레인을 나타낸 것이다(XYL1 대 GRE3).
도 53 은 40 g/L 의 자일로스에서 성장한 유전자 조작된 S. 세레비시아 에 의한 자일로스 발현에서 과잉-발현된(over-expressing) XYL1 대 GRE3 의 효과를 나타낸 것이다.
도 54 은 80 g/L 의 자일로스에서 성장한 유전자 조작된 S. 세레비시아 에 의한 자일로스 발현에서 과잉-발현된(over-expressing) XYL1 대 GRE3 의 효과를 나타낸 것이다.
도 55 는 상이한 야생형 LAD 효소의 열 특성(thermal property) 및 pH-의존적인 특성(pH-dependent properties)을 나타낸 것이다 : LAD(■), tlLAD(◆) 및 pcLAD(●). (A) 온도-의존적인 촉매 활성도(Temperature-dependent catalytic activities), (B) 오버타임(over time) 동안의 50 ℃ 에서의 열의 불활성화(Thermal inactivation) 및 (C) pH-의존적인 촉매 활성도. 에러바(Error bars)는 평균의 표준 오차(standard error)를 나타낸다(n = 3).
도 56 은 N. 크라사 및 P. 스티피티스(psXDH)로부터의 XDH 의 정렬(alignment)을 나타낸 것이다.
도 57 은 N. 크라사 LAD 및 XDH 의 pH 비율 프로파일(pH rate profiles)의 비교를 나타낸 것이다. LAD 의 특성으로부터의 획득한 데이터는 일반적인 완충용액 MES/트리스/글리신에서 수행하였고, 낮은 pH 값에 대해 일반적인 완충용액 아세트산/MES/트리스에서 수행한 ncXDH[폐쇄된 삼각형(closed triangles)] 및 ncLAD[폐쇄된 원형(closed circles)] 에 대한 데이터는 중복되었다.
도 58 은 박테리오드 스테코리스(Bacteroids stercoris , BtXI), 비피도박테리움 롱검(Bifidobacterium longum , BfXI) 및 코돈-최적화된 BtXI(codon-optimized BtXI)에 대한 BtXIO 로부터의 자일로스 이성질화효소(xylose isomerase enzyme)를 갖는 형질전환된(transformed) S. 세레비시아 스트레인 L2612 에 의한 에탄올 생산을 나타낸 것이다. XI 유전자는 pRs424TEF 벡터 내로 클론되었다(clone).
도 59 는 벡터 pRS403TEF 에 의해 이의 게놈(genome) 내로 통합된(integrated) BtXI 를 갖는, S. 세레비시아 스트레인 D452-2 에 의한 자일로스 소비 및 에탄올 생산을 나타낸 것이다. 플라스미드로부터 BtXI 가 발현된 S. 세레비시아 스트레인 L2612 에 의한 자일로스-발효와 비교하였다.
도 60 은 통합된 BtXI 및 발현된 XYL2 또는 XYL3 또는 XYL2 및 XYL3 을 함유하는 S. 세레비시아 스트레인에 의한 자일로스 발효를 나타낸 것이다.
도 61 은 자일로스의 효율적인 대사작용을 위해 오탄당 인산경로(pentose phosphate pathway)에서 포함된 GND1 과 같은 과잉-발현된 효소를 유전자 조작한 S. 세레비시아 에서 XYL3 발현의 필요성을 나타낸 것이다.
도 62 는 발효 파라미터(fermentation parameters)에서 대장균, S. 세레비시아 및 P. 스티피티스에서 NCU09705 상동성의 과잉-발현의 효과를 나타낸 것이다. (A) 셀로비오스 소비, 성장 및 에탄올 생산 ; 및 (B) 에탄올 수득률 및 생산성에서 galM, GALIO-Sc, GALIO-Ps, YHR210C 및 YNR071C 의 과잉-발현.
도 63 은 곰팡이(N. 크라사)로부터의 셀로덱스트린 동화 경로(assimilation pathway) 및 S. 세레비시아 내의 자일로스-발효 효모 P. 스티피티스로부터의 변형된 자일로스 대사경로의 통합을 통해서 글루코스 리프레션(repression)없이 셀로비오스 및 자일로스의 동시적인 공동-발효(co-fermentation)를 가능하게 하는 실험 설계를 나타낸 것이다. (A) 두 개의 대사작용이 가능하지 않는 당(non-metabolizable sugars)(셀로비오스 및 자일로스)을 발효시킬 수 있는 효모 스트레인을 유전자 조작하기 위한 스트레인 개선 전략(strain improvement strategy). 셀로덱스트린 동화 경로는 N. 크라사로부터의 셀로덱스트린 운반체(NCU00801) 및 세포 내의 β-글루코시다아제(NCU00130)으로 이루어져 있다. 변형된 자일로스 대사 경로는 자일로스 환원효소 동질효소(xylose reductase isozymes)(야생형 XR 및 돌연변이체 XR R.276H), 자일리톨 탈수소효소(XYL2) 및 자일루로키나아제(xylulokinase)(XKS1)를 사용하였다. (B) 유전자 조작된 S. 세레비시아에 의한 글루코스 및 자일로스를 함유하는 당 혼합물의 발효 프로파일(Fermentation profile)은 이러한 연구에서 진전되었다(develop). 글루코스가 고갈된 후에만 자일로스 발효가 시작되기 때문에 글루코스 발효는 자일로스 발효를 억제한다. (C) 유전자 조작된 S. 세레비시아에 의한 셀로비오스 및 자일로스를 함유하는 당 혼합물의 발효 프로파일은 이러한 실험에서 진전되었다. 탄소원(carbon source)이 다른 물질의 소비를 억제하지 않음에 따라 셀로비오스 및 자일로스는 동시에 사용되었다.
도 64 는 플라스미드 구성(plasmid construction)에 대한 도식(scheme)을 타나낸 것이다. pRS425 셔틀벡터(shuttle vector)는 DNA 조립 방법(DNA assembler method)(Shao 등의, 2009)를 사용한 셀로비오스 운반체 및 β-글루코시다아제 유전자를 조립한 다음에 선 모양으로 만들었다(linearized).
도 65 는 시간의 함수(function)으로서의 S. 세레비시아 스트레인 (A) SL01, (B) SL04, (C) SL02, (D) SL05, (E) SL03, (F) SL06 및 (G) SL00 에 의한 4 % 셀로비오스 및 5 % D-자일로스의 공동-발효 동안의 셀로비오스(■), 글루코스(●), D-자일로스(▲), 에탄올(▼), 및 바이오매스(□)의 농도의 변화를 나타낸 것이다.
도 66 은 시간의 함수로서 도표로 나타낸, 진탕-플라스크(a,b) 또는 바이오리액터(bioreactors)(c, d)에서 셀로비오스-자일로스 혼합물에서 성장한 S. 세레비시아 스트레인 SL01(a, b) 및 SL00(b, d)에서의 셀로비오스(■), 글루코스(●), D-자일로스(▲), 에탄올(▼), 및 바이오매스(□)의 농도의 변화를 나타낸 것이다.
도 67 은 시간의 함수로서 도표로 나타낸, 바이오리액터에서 5 g/L 글루코스-40 g/L 셀로비오스-50 g/L 자일로스 혼합물(a, b) 또는 10 g/L 글루코스-40 g/L 셀로비오스-50 g/L 자일로스 혼합물(c, d)를 함유하는 배지에서 성장한 S. 세레비사이 스트레인 SL01(a, c) 및 SL00(b, d)에서의 셀로비오스(■), 글루코스(●), D-자일로스(▲), 에탄올(▼), 및 바이오매스(□)의 농도의 변화를 나타낸 것이다.
도 68 은 (A) NCU00801, (B) NCU00809 및 (C) NCU08114 가 발현된 β-글루코시다아제(NCU00130)-함유 S. 세레비시아 스트레인에 의한 셀로비오스 활용의 비교를 나타낸 것이다. 부호 : 셀로비오스(■), 에탄올(◆) 및 OD600(●).
도 69 는 두 가지의 당의 다양한 농도를 함유하는 혼합물에서 성장한 S. 세레비시아 스트레인 DA24-16BT3 에 의한 셀로비오스 및 자일로스의 공동-발효를 나타낸 것이다 : (A) 20 g/L(각각)의 셀로비오스 및 자일로스, (B) 30 g/L(각각)의 셀로비오스 및 자일로스, 및 (C) 40 g/L(각각)의 셀로비오스 및 자일로스. 부호 : 셀로비오스(▲), 자일로스(■), 에탄올(◆) 및 OD600(●).
도 70 은 S. 세레비시아 스트레인 DA24-16BT3 에 의한 셀로비오스 및 자일로스의 공동-발효의 상승효과(synergistic effect)를 나타낸 것이다. 부호 : 셀로비오스(▲), 자일로스(■), 에탄올(◆) 및 OD600(●) : (A) 40 g/L 셀로비오스, (B) 40 g/L(각각)의 셀로비오스 및 자일로스, 및 (C) 40 g/L 자일로스.
도 71 은 S. 세레비시아 스트레인 DA24-16BT3 및 야생형 P. 스티피티스 스트레인에 의한 글루코스, 셀로비오스 및 자일로스의 공동-발효를 나타낸 것이다. 부호 : 셀로비오스(▲), 자일로스(■), 에탄올(◆), OD600(●) 및 글루코스(▼). (A) DA24-16BT3 및 (B) P. 스티피티스.
도 72 는 S. 세레비시아 스트레인 DA24-16BT3 에 의한 셀로비오스 및 자일로스의 c-발효 동안 셀로트리오스 및 셀로테트라오스 축적(accumulation)을 시사하는, 각각의 시점으로부터의 HPLC 크로마토그램(HPLC chromatograms)을 나타낸 것이다.
도 73 은 셀로비오스 및 자일로스의 공동-발효 동안의 S. 세레비시아 스트레인 DA24-16BT3 에 의한 22 시간 발효 후의 발효 배지에서 셀로덱스트린 축적을 보여주는 HPAEC 분석을 나타낸 것이다. [Gl : 클루코스, G2 : 셀로비오스, G3 : 셀로트리오스, G4 : 셀로테트라오스, 및 G5 : 셀로펜타오스(cellopentaose)].
도 74 는 40 g/L(각각)의 셀로비오스 및 자일로스의 공동-발효 동안에, 다중-복제 플라스미드(multi-copy plasmid)에서의 (A) NCU00801 의 통합된 복제(integrated copy) 및 (B) NCU00801 이 발현된 S. 세레비시아 형질전화체(S. cerevisiae transformants)에 의한 당 이용의 비교를 나타낸 것이다. 부호 : 셀로비오스(▲), 자일로스(■), 에탄올(◆), 및 OD600(●).
도 75 는 (A) 연구에서 사용된 두 가지 상이한 S. 세레비시아 스트레인 : DA24-16 및 D452BT, 두 가지 상이한 효모 스트레인의 배양(cultivation)에 의한 에탄올 생산을 나타낸 것이다. 자일로스 분자는 오각형으로 나타내었고 셀로비오스 분자는 두 개의 육각형으로서 나타내었다 ; 및 (B) 자일로스-발효 스트레인 및 셀로비오스-발효 스트레인의 혼합된 배양물(Mixed cultures).
도 76 은 N. 크라사에서 354 자일란-유도 유전자(354 xylan-induced genes)의 목록을 나타낸 것이다.
도 77 은 다양한 N. 크라사 녹-아웃 스트레인(knock-out strains)에 대한 분비된 단백질 수준(secreted protein levels), 감소된 당(reducing sugar) 및 아조-자일라나아제 활성도(azo-xylanase activity)를 나타낸 것이다. 분비된 단백질 수준은 모든 스트레인에 대해서 상대적인 상수(relatively constant)이다.
도 78 은 (A) 야생형, △NCU05137 및 △NCU05137/△NCU05137-GFP 네우로스포라 스트레인에 대한 총 분비된 단백질 및 CMC-활성도, 및 (B) 세 가지 상이한 스트레인의 배양물(cultures)의 상청액(supernatant)에서의 총 단백질의 쿠마시 염색(Coomassie strain)을 나타낸 것이다.
도 79 는 분생자(conidia)에서 NCU05137-GFP 의 위치(localization)를 나타낸 것이다.
도 80 은 균사 팁(hypha tip)에서 NCU05137-GFP 의 위치를 나타낸 것이다.
발명의 상세한 설명
본 발명은, 트랜스맴브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11 및 α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포에 관한 것이되, 하기의 하나 또는 그 이상은 사실이다(true) : 트랜스맴브레인 α-헬릭스 1 은 SEQ ID NO: 1 을 포함하고, 트랜스맴브레인 α-헬릭스 2 는 SEQ ID NO: 2 를 포함하고, the loop connecting 트랜스맴브레인 α-헬릭스 2 와 트랜스맴브레인 α-헬릭스 3 를 연결하는 루프는 SEQ ID NO: 3 을 포함하고, 트랜스맴브레인 α-헬릭스 5 는 SEQ ID NO: 4 를 포함하고, 트랜스맴브레인 α-헬릭스 6 은 SEQ ID NO: 5 를 포함하고, 트랜스맴브레인 α-헬릭스 6 과 트랜스맴브레인 α-헬릭스 7 사이의 서열은 SEQ ID NO: 6 을 포함하고, 트랜스맴브레인 α-헬릭스 7 은 SEQ ID NO: 7 를 포함하고, 및 트랜스맴브레인 α-헬릭스 10 및 트랜스맴브레인 α-헬릭스 11 및 이들 사이의 서열은 SEQ ID NO: 8 을 포함하고, 및 상기 폴리펩티드는 세포 내로의 셀로덱스트린을 수송한다. 본원은, 세포 내로 셀로덱스트린의 수송을 증가시키는 방법, 셀로덱스트린을 포함하는 배지에 세포의 성장을 증가시키는 방법, 셀룰로스-유도 및 헤미셀룰로스-유도 당을 공동-발효시키는 방법 및 숙주 세포를 이용한 탄화수소 또는 탄화수소 유도체를 생성하는 방법을 추가적으로 기재하고 있다. 본원은, 세포 내로 오탄당을 수송하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포, 세포 내로 오탄당의 수송을 증가시키는 방법, 오탄당 당을 포함하는 배지에서 세포의 성장을 증가시키는 방법, 및 세포 내로 오탄당을 수송하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하는 탄화수소 또는 탄화수소 유도체를 생성하는 방법을 추가적으로 기재하고 있다.
본원에서 사용된 바와 같이, 셀로덱스트린은 다양한 길이(varying length)의 글루코스 중합체를 나타내고, 이로 제한되지 않는, 셀로비오스(2 글루코스 단량체), 셀로트리오스(cellotriose)(3 글루코스 단량체), 셀로테트라오스(cellotetraose)(4 글루코스 단량체), 셀로펜타오스(cellopentaose)(5 글루코스 단량체) 및 셀로헥사오스(cellohexaose)(6 글루코스 단량체)를 포함한다.
본원에서 사용된 바와 같이, 당은 단당류(monosaccharides)[예를 들어, 글루코스, 과당(fructose), 갈락토스, 자일로스, 아라비노스], 이당류(예를 들어, 셀로비오스, 수크로스, 락토오스, 말토오스), 및 올리고당(일반적으로 3 내지 10 성분(component)의 단당류)을 나타낸다.
본 발명의 폴리뉴클레오티드
본원의 본 발명은 숙주 세포 및 이러한 숙주 세포를 이용한 방법에 관한 것이되, 상기 숙주 세포는 다양한 당을 수송할 수 있는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함한다.
본원에서 사용된 바와 같이, 용어 "폴리뉴클레오티드", "핵산 서열", "핵산의 서열" 및 이의 변형은 폴리데옥시리보뉴클레오티드(2-데옥시-D-리보오스를 포함), 폴리리보뉴클레오티드(D-리보오스를 포함), 퓨린 또는 피리미딘 염기의 N-글리코시드인 기타 다른 종류의 폴리뉴클레오티드, 및 DNA 및 RNA 에서 발견된 바와 같이, 상기 중합체가 염기 짝짓기(base pairing) 및 염기중첩(base stacking)을 가능하게 하는 입체배치(configuration)에서의 핵염기(nucleobases)를 포함한다면, 비-뉴클레오티드 백본(non-nucleotidic backbones)을 포함하는 그 밖의 중합체를 총칭한다. 따라서, 이러한 용어는 알려진 종류의 핵산 서열 변형물, 예를 들어 유사물(analog)을 갖는 하나 또는 그 이상의 자연적으로 발생하는 뉴클레오티드의 치환 ; 예를 들어, 전하되지 않은 결합(uncharged linkages)을 갖는 것들[예를 들어, 메틸 포스페이트(methyl phosphonates), 포스포트리에스테르(phosphotriesters), 포스포르아미데이트(phosphoramidates), 카르바메이트(carbamates) 등], 음전화된 결합(negatively charged linkages)을 갖는 것들[예를 들어, 포스포로티오에이트(phosphorothioates), 포스포로디티오에이트(phosphorodithioates) 등], 및 양전화된 결합(positively charged linkages)을 갖는 것들[예를 들어, 아미노알킬포스포르아미데이트(aminoalkylphosphoramidates), 아미노알킬포스포트리에스테르(aminoalkylphosphotriesters) 등]와 같은 인터-뉴클레오티드 변형물(inter-nucleotide modifications) ; 예를 들어 단백질[뉴클레아제(nucleases), 독소(toxins), 항체, 단일 펩티드, 폴리-L-리신 등을 포함)과 같은 펜던트의 모이어티(pendant moieties)를 포함하는 것들 ; 인터킬레이터(intercalators)를 갖는 것들[예를 들어 아크리딘(acridine), 소랄렌(psoralen) 등] 및 킬레이터를 포함하는 것들[예를 들어, 금속, 방사성 금속, 붕소, 산화금속(oxidative metals) 등]을 포함한다. 본원에 사용된 바와 같이, 뉴클레오티드 및 폴리뉴클레오티드에 대한 부호(symbols)는 IUPAC-IUB Commission of Biochemical Nomenclature 에 의해 권장된 것이다(Biochem. 9:4022, 1970).
본원에서 사용된 바와 같이, "폴리펩티드"는 다수의 연이은 중합 아미노산 잔기(a plurality of consecutive polymerized amino acid residues)(예를 들어, 적어도 약 15 가지의 연이은 중합 아미노산 잔기, 선택적으로 적어도 약 30 가지의 연이은 중합 아미노산 잔기, 적어도 약 50 가지의 연이은 중합 아미노산 잔기)를 포함하는 아미노산 서열이다. 많은 예에서, 폴리펩티드는, 운반체, 전사인자(transcription factor), 알려지지 않은 기능(unknown function)의 예측 단백질(predicted protein), 또는 이의 도메인 또는 단백질 또는 단편인 중합 아미노산 잔기 서열(polymerized amino acid residue sequence)을 포함한다. 운반체는 이온, 작은 분자, 또는 생체막(biological membrane)을 가로지르는 탄수화물과 같은 고분자(macromolecules)를 포함하는 것이다. 전사인자는 유전자 발현을 조절할 수 있고, 숙주 세포에서 유전자 발현을 증가시키거나 감소시킬 수 있다. 폴리펩티드는, 선택적으로 변형된 아미노산 잔기(modified amino acid residues), 코돈(codon)에 의해 코드되지 않는 자연적으로 발생한 아미노산 잔기(naturally occurring amino acid residues) 및 비-자연적으로 발생한 아미노산 잔기를 포함한다.
본원에서 사용된 바와 같이, "단백질" 은 아미노산 서열, 올리고펩티드, 펩티드, 폴리펩티드 또는 자연적으로 발생하거나 합성된(naturally occurring or synthetic) 이의 부분(portions)에 관한 것이다.
본 발명의 재조합 폴리뉴클레오티드는 표 10, Supplemental Data, Dataset SI, page 3 in Tian et al., 2009 ; 표 14, 15, 16, 29 ; 또는 도 76 에 나타낸 유전자 중 어떠한 것에 의해 코드되는 폴리펩티드를 코드하는 어떠한 폴리뉴클레오티드를 포함한다. 바람직한 실시형태에서, 본 발명의 폴리뉴클레오티드는, 서열 NCU00801, NCU00809, NCU08114, NCU00130, NCU00821, NCU04963, NCU06138, STL12/XUT6, SUT2, SUT3, XUT1, XUT3, NCU07705, NCU05137, NCU01517, NCU09133, or NCU10040 중 어떤 것에 의해 코드되는 폴리펩티드를 코드하는 어떠한 폴리뉴클레오티드를 포함한다.
[표 1]
특정 실시형태에서, 본 발명의 재조합 폴리뉴클레오티드는, 표 10, Supplemental Data, Dataset SI, page 3 in Tian et al., 2009 ; 표 14, 15, 16, 29 ; 또는 도 76 에 나타낸 유전자 중 어떠한 것에 의해 코딩되는 폴리펩티드와 적어도 약 20 %, 적어도 약 29 %, 적어도 약 30 %, 적어도 약 40 %, 적어도 약 50 %, 적어도 약 55 %, 적어도 약 60 %, 적어도 약 65 %, 적어도 약 70 %, 적어도 약 75 %, 적어도 약 80 %, 적어도 약 85 %, 적어도 약 90 %, 적어도 약 92 %, 적어도 약 94 %, 적어도 약 96 %, 적어도 약 98 %, 적어도 약 99 % 또는 적어도 약 100 % 의 아미노산 동일성 잔기 서열(amino acid residue sequence)을 갖는 폴리펩티드를 코드한다. 바람직한 실시형태에서, 본 발명의 폴리뉴클레오티드는, 서열 NCU00801, NCU00809, NCU08114, NCU00130, NCU00821, NCU04963, NCU06138, STL12/XUT6, SUT2, SUT3, XUT1, XUT3, NCU07705, NCU05137, NCU01517, NCU09133 또는 NCU10040 중 어떠한 것에 의해 코드되는 폴리펩티드와 적어도 약 20 %, 적어도 약 29 %, 적어도 약 30 %, 적어도 약 40 %, 적어도 약 50 %, 적어도 약 55 %, 적어도 약 60 %, 적어도 약 65 %, 적어도 약 70 %, 적어도 약 75 %, 적어도 약 80 %, 적어도 약 85 %, 적어도 약 90 %, 적어도 약 92 %, 적어도 약 94 %, 적어도 약 96 %, 적어도 약 98 %, 적어도 약 99 % 또는 적어도 약 100 % 의 아미노산 동일성 잔기 서열을 갖는 폴리펩티드를 코드한다.
본 발명의 폴리뉴클레오티드는 상기 나타낸 유전자에 의해 코드되는 폴리펩티드의 보존적으로 변형된 변이체(conservatively modified variants)를 코드하는 폴리뉴클레오티드를 추가로 포함한다. 본원에 사용된 바와 같은 "보존적으로 변형된 변이체(Conservatively modified variants)" 는 화학적으로 유사한 아미노산을 갖는 아미노산의 치환을 야기하는, 폴리펩티드 서열에 개별적인 치환(substitutions), 결실(deletions) 또는 첨가를 포함한다. 기능적으로 유사한 아미노산을 제공하는 보존적 치환 표(Conservative substitution tables)는 본 분야에서 널리 공지되어 있다. 이러한 보존적으로 변형된 변이는 다형성 변이체(polymorphic variants), 이종간의 상동체(interspecies homologs) 및 디스클로우저(disclosure)의 대립유전자(alleles)를 제외하지 않고 이를 포함한다. 하기의 8 가지 그룹은 서로에 대한 보존적 치환인 아미노산을 포함한다 : 1) 알라닌(A), 글리신(G); 2) 아스파르트산(D), 글루탐산(E); 3) 아스파라진(N), 글루타민(Q); 4) 아르기닌(R), 리신(K); 5) 이소류신(I), 류신(L), 메티오닌(M), 발린(V); 6) 페닐알라닌(F), 티로신(Y), 트립토판(W); 7) 세린(S), 트레오닌(T); 및 8) 시스테인(C), 메티오닌(M)[예를 들어 Creighton, Proteins(1984) 참조].
본 발명의 폴리뉴클레오티드는, 표 10, Supplemental Data, Dataset SI, page 3 in Tian et al., 2009 ; 표 14, 15, 16, 29 ; 또는 도 76 에 나타낸 유전자중 어떠한 것에 의해 코드되는 폴리펩티드의 오솔로그(orthologs) 또는 상동체(homologs)를 코드하는 폴리뉴클레오티드를 추가로 포함한다. 본원에 사용된 바와 같은 "상동체(Homology)" 는 참조서열(reference sequence) 및 두 번째 서열의 적어도 하나의 단편 사이의 서열 유사성(sequence similarity)에 관한 것이다. 상동체는 본 분야에서 공지된 어떠한 방법, 바람직하게 참조 서열과 단일 두 번째 서열(single second sequence) 또는 서열의 단편, 또는 서열의 데이터베이스와 비교하는 BLAST 도구(BLAST tool)를 사용하여 확인할 수 있다. 하기에 기재된 바와 같이, BLAST 는 퍼센트 동일성(identity) 및 유사성(similarity)을 기반으로 한 서열과 비교할 수 있다. 본원에서 사용된 바와 같은, "오솔로그(Orthology)"는 공통 조상 유전자(common ancestor gene)로부터 유래된 상이한 종(different species )에서의 유전자에 관한 것이다.
두 가지 또는 그 이상의 핵산 또는 폴리펩티드 서열의 맥락에서 용어 "동일한(identical)" 또는 퍼센트 "동일성(identity)"은 동일한 두 가지 또는 그 이상의 서열 또는 서브서열(subsequences)에 관한 것이다. 비교 윈도우(comparison window), 또는 다음의 서열 비교 알고리즘 또는 매뉴얼 정렬(manual alignment)을 사용하거나 또는 육안검사(visual inspection)에 의해 측정된 바와 같은 지정된 영역(designated region) 상에서 최대한의 관련성(maximum correspondence)에 대해서 비교하고 정렬하였을 때, 만약 두 가지 서열이 의 아미노산 동일성 잔기 또는 뉴클레오티드의 명시된 퍼센트(예를 들어, 명시된 영역, 또는 명시되지 않은 전체 서열 상에서 29 % 동일성, 선택적으로 30 %, 40 %, 50 %, 55 %, 60 %, 65 %, 70 %, 75 %, 80 %, 85 %, 90 %, 95 %, 99 % 또는 100 % 동일성)를 갖는다면, 두 서열은 "상당히 동일하다(substantially identical)". 선택적으로, 동일성은, 길이(length)에서 적어도 약 50 개의 뉴클레오티드(또는 10 개의 아미노산)인 영역, 또는 보다 바람직하게 길이에서 100 내지 500 개 또는 1000 개 또는 그 이상의 뉴클레오티드(또는 20, 50, 200 개 또는 그 이상의 아미노산)인 영역 상에서 존재한다.
서열 비교(sequence comparison)에서, 일반적으로 하나의 서열은, 실험 서열과 비교하기 위해, 참조 서열(reference sequence)로서 작용한다. 서열 비교 알고리즘(sequence comparison algorithm)을 사용할 때, 실험 및 참조 서열을 컴퓨터에 입력하고, 만약 필요하다면 서열 코디네이트(subsequence coordinates)를 지정하고, 서열 알고리즘 프로그램 파라미터(sequence algorithm program parameters)를 지정한다. 디폴트 프로그램 파라미터(Default program parameters)를 사용할 수 있고, 또는 선택적인 파라미터를 지정할 수 있다. 그리고 난 다음에 서열 비교 알고리즘은, 프로그램 파라미터를 기반으로 한 참조 서열에 대한 상대적인 테스트 서열에 대한 퍼센터 서열 동일성(percent sequence identities)을 계산한다. 동일성에 대해서 두 가지 서열을 비교하였을 때, 서열이 근접할 필요는 없지만, 갭(gap)은 전체적인 퍼센트 동일성을 감소시킬 수 있는 페널티(penalty)을 줄 수 있다. blastn 에 대해서, 디폴트 파라미터(default parameters)는 갭 오프닝 페널티(Gap opening penalty) = 5 및 갭 오프닝 페널티 = 2 이다. blastp 에 대해서, 디폴트 파라미터는 갭 오프닝 페널티 = 11 및 갭 익스텐션 페널티(Gap extension penalty) = 1 이다.
본원에 사용된 바와 같은 "비교 윈도우(comparison window)"는 인접한 위치의 수 중 어떠한 것의 부분(segment)과 관련된 것을 포함하지만, 서열은 두 가지의 서열이 최적으로 정렬된 후에 인접한 위치의 동일한 번호의 참조서열과 비교하여, 20 내지 60, 일반적으로 약 50 내지 약 200, 보다 일반적으로 약 100 내지 약 150 로 제한되지 않는다. 비교를 위한 서열 정렬의 방법은 본 분야에 공지되어 있다. 비교를 위한 서열의 최적의 정렬은, Smith and Waterman (1981)의 일부의 상동성 알고리즘(local homology algorithm), Needleman and Wunsch (1970) J Mol Biol 48(3):443-453 의 상동성 정렬 알고리즘(homology alignment algorithm), Pearson and Lipman (1988) Proc Natl Acad Sci USA 85(8):2444-2448 의 유사성 방법에 대한 검색, 이러한 알고리즘(GAP, BESTFIT, FASTA, and TFASTA in the Wisconsin Genetics Software Package, Genetics Computer Group, 575 Science Dr., Madison, WI)의 컴퓨터화된 이행(computerized implementations) 또는 메뉴얼 정렬 및 육안 검사[예를 들어, Brent et al ., (2003) Current Protocols in Molecular Biology, John Wiley & Sons, Inc. (Ringbou Ed) 참조]에 의해 할 수 있다.
퍼센트 서열 동일성 및 서열 유사성에 대한 적절한 알고리즘의 두 가지 예는, 각각 Altschul 등의. (1997) Nucleic Acids Res 25(17):3389-3402 및 Altschul 등의. (1990) J. Mol Biol 215(3)-403-410 에 기재된 BLAST 및 BLAST 2.0 알고리즘이다. BLAST 분석을 수행하기 위한 소프트웨어는 National Center for Biotechnology Information 을 통해 공개적으로 입수가능하다. 이러한 알고리즘은 데이타베이스 서열에서 동일한 길이의 마디(word)와 함께 정렬하였을 때, 몇몇 포지티브-값의 한계 점수 T (positive-valued threshold score T)를 맞추거나 또는 충족시키는, 질의 서열(query sequence)에서 길이 W 의 짧은 단어(short words)를 확인하여 첫 번째로 확인된 높은 점수의 서열 짝(high scoring sequence pairs, HSPs)을 포함한다. T 는 인접한 단어 점수 한계(neighborhood word score threshold)로서 언급된 것이다(Altschul et al., supra). 이러한 초기의 인접한 단어 히트(initial neighborhood word hits)는 이들을 포함하는 보다 긴 HSPs 를 발견하기 위해 초기 검색에 대한 시드(seeds)로서의 역할을 한다. 단어 히트(word hits)는 누적된 정렬 점수(cumulative alignment score)가 증가될 수 있을 때까지 각각의 서열을 따라 양 방향(both directions)으로 연장되었다. 누적된 점수는 뉴클레오티드 서열, 파라미터 M[어울리는 잔기(matching residues)의 짝(pair)에 대한 보상 점수(reward score) ; 항상 > 0] 및 N[어울리지 않는 잔기(mismatching residues)에 대한 페널티 점수(penalty score) ; 항상 < 0]에 대해 사용되어 계산되었다. 아미노산 서열에서, 스코링 매트릭스(scoring matrix)는 누적된 점수를 계산하기 위해 사용되었다. 각각의 방향에서 단어 히트의 연장(Extension)은, 누적된 정렬 점수(cumulative alignment score)가 이의 최대한의 성취된 값(achieved value)으로부터의 다수의 X(quantity X)에 의해 떨어졌을 때 ; 누적된 점수가, 하나 또는 그 이상의 네가티브-스코링 잔기 정렬(negative-scoring residue alignments)의 축적으로 인하여 0 이하로 갔을 때 ; 또는 어느 하나의 서열이 말단으로 도달하였을 때에 정지한다. BLAST 알고리즘 파라미터 W, T 및 X 은 민감성(sensitivity) 및 정렬의 속도를 결정한다. BLASTN 프로그램(뉴클레오티드 서열에 대한)은 디폴트(defaults) 11 의 워드랭스(wordlength)(W), 기대값(expectation)(E) 또는 10, M=5, N=-4, 및 둘 다의 가닥의 비교로서 사용되었다. 아미노산 서열에 대해서, BLASTP 프로그램은 디폴트 3 의 워드랭스(W), 10 의 기대값(E) 및 BLOSUM62 스코링 매트릭스[Henikoff and Henikoff, (1992) Proc Natl Acad Sci USA 89(22):10915-10919 참조], 50 의 정렬(alignments)(B), 10 의 기대값, M=5, N=-4 및 둘 다의 가닥의 비교로서 사용되었다.
BLAST 알고리즘은 또한 두 가지 서열 사이의 유사성의 통계적인 분석(statistical analysis)을 위해 또한 실시하였다(예를 들어, Karlin and Altschul, (1993) Proc Natl Acad Sci USA 90(12):5873-5877 참조). BLAST 알고리즘에 의해 제공된 유사성의 하나의 척도(measure)는, 두 가지의 뉴클레오티드 또는 아미노산 서열 사이의 매치(match)가 우연히 발생할 수 있는 가능성의 표시(indication)를 제공하는, 가장 작은 합계 확률(sum probability)(P(N))이다. 예를 들어, 만약 테스트 핵산과 참조 핵산의 비교에서 가장 작은 합계 확률이 약 0.2 미만, 보다 바람직하게 약 0.01 미만 및 가장 바람직하게 약 0.001 미만이라면, 핵산은 참조서열에 유사한 것으로 간주된다.
상기에 나타낸 서열 동일성 퍼센트 이외의, 두 가지 핵산 서열 또는 폴리펩티드가 상당히 동일한 또 다른 표시(indication)는, 하기에 나타낸 바와 같이, 첫 번째 핵산에 의해 코드된 폴리펩티드가 두 번째 핵산에 의해 코드된 폴리펩티드에 대하여 증가된 항체와 면역학적으로 교차-반응성(immunologically cross-reactive)을 갖는 경우이다. 따라서, 예를 들어 두 가지 펩티드가 보존적 치환에 의해서만 다른 경우에, 폴리펩티드는 두 번째 폴리펩티드와 일반적으로 상당히 동일하다. 두 가지 핵산 서열이 상당히 동일한 또 다른 표시는, 하기에 나타낸 바와 같이, 두 가지 분자 또는 이들의 보체(complements)가 엄격한 조건(stringent conditions) 하에서 서로가 혼성화(hybridize)되는 경우이다. 두 가지 핵산 서열이 상당히 동일한 또 다른 표시는, 동일한 프라이머를 서열을 증폭시키기 위해 사용할 수 있는 경우이다.
본원에 나타낸 바와 같이, 본 발명의 폴리뉴클레오티드는, NCU00988, NCU10021, NCU04963, NCU06138, NCU00801, NCU08114 및 NCU05853 를 포함하는 주요한 촉진 상과 당 운반체 패밀리(Major Facilitator Superfamily sugar transporter family)의 멤버(members)를 포함한다. 운반체의 주요한 촉진 상과(Major Facilitator Superfamily, MFS)(운반체 유형 # 2.A.1)의 멤버(Members)는, 세포내 N- 및 C-말단을 갖는 12 가지의 트랜스멤브레인 α-헬릭스로 항상 구성되어있고[S. S. Pao, I. T. Paulsen, M. H. Saier, Jr., Microbiol Mol Biol Rev 62, 1 (Mar, 1998)], 반면에 MFS 운반체의 일차 서열(primary sequence)은 폭넓게 다양하고, 모두는 E. coli 락토오스 투과효소(lactose permease)(LacY)[J. Abramson et al ., Science 301, 610 (Aug 1, 2003)] 및 E. coli Pi/글리세롤-3-포스페이트(GlpT)[Y. Huang, M. J. Lemieux, J. Song, M. Auer, D. N. Wang, Science 301, 616 (Aug 1, 2003)]의 삼차구조를 공유한다. 이러한 예에서, 6 가지의 N- 및 C- 말단 헬릭스는 헬릭스 6 및 7 사이의 긴 세포질 루프(long cytoplasmic loop)에 의해 연결된 두 가지의 별개의 도메인을 형성한다. 이러한 대칭(symmetry)은 MFS 에 대해 주어진 증가(given rise)를 갖는 것을 생각하는 복제 사건(duplication event)에 해당한다. 친수성 캐비티(hydrophilic cavity) 내의 기질 결합은 N-말단 도메인의 헬릭스 1, 2, 4 및 5, 및 C-말단 도메인의 헬릭스 7, 8, 10 및 11 에 의해 형성된다. 이러한 캐비티는 헬릭스 3, 6, 9 및 12 에 의해 안정된다.
MFS 의 당 운반체 패밀리(운반체 분류 # 2.A.1.1)는 트랜스멤브레인 헬릭스 6 및 12[PESPR (SEQ ID NO: 9)/PETK (SEQ ID NO: 10)], 및 루프 2 및 8(GRR/GRK)[M. C. Maiden, E. O. Davis, S. A. Baldwin, D. C. Moore, P. J. Henderson, Nature 325, 641 (Feb 12-18, 1987)]에서 발견된 모티프(motifs)로 규정된다. 이러한 패밀리에 대해서 전체적인 Hidden Markov Model(HMM)는 pfam.janelia.org/family/PF00083#tabview=tab3 에서 볼 수 있다. PROSITE[N. Hulo et al., Nucleic Acids Res 34, D227 (Jan 1, 2006)]은 이러한 패밀리의 멤버를 확인하기 위해 두 가지의 모티프를 사용하였다. 첫 번째는 [LIVMSTAG] - [LIVMFSAG] - {SH} - {RDE} - [LIVMSA] - [DE] - {TD} - [LIVMFYWA] - G - R - [RK] - x(4,6) - [GSTA](SEQ ID NO: 11)이다. 두 번째는 [LIVMF] - x - G - [LIVMFA] - {V} - x - G - {KP} - x(7) - [LIFY] - x(2) - [EQ] - x(6) - [RK] (SEQ ID NO: 12)이다. PROSITE 모티프를 해독하는 방법을 예로서 하기의 모티프 [AC]-x-V-x(4)-{ED}는 [Ala 또는 Cys]-어떠한 것(any)-Val-어떠한 것-어떠한 것-어떠한 것-어떠한 것-{Glu 또는 Asp 외의 어떠한 것}({any but Glu or Asp}) (SEQ ID NO: 13)로서 번역된다.
본원에 기재된 바와 같이, NCU00801, NCU00809, NCU08114, XP_001268541.1 및 LAC2 은 셀로덱스트린을 수송하는 폴리펩티드를 코드하는 것으로 발견되었다. 게다가, 알라닌 스캐닝 실험 및 서열 분석은 12 가지의 트랜스멤브레인 α-헬릭스를 포함하는 재조합 폴리펩티드를 결정하기 위해 사용되었고, SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, and SEQ ID NO: 8 으로 이루어진 하나 또는 그 이상의 서열은 셀로덱스트린을 수송하는 폴리펩티드를 코드한다.
따라서, 하나의 양상에서, 본 발명은 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 제공하고, 트랜스멤브레인 α-헬릭스 1 은 SEQ ID NO: 1 을 포함한다. 또 다른 양상에서, 본 발명은 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 제공하고, 트랜스멤브레인 α-헬릭스 2 는 SEQ ID NO: 2 를 포함한다. 또 다른 양상에서, 본 발명은 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 제공하고, 트랜스멤브레인 α-헬릭스 2 및 트랜스멤브레인 α-헬릭스 3 을 연결하는 루프는 SEQ ID NO: 3 을 포함한다. 또 다른 양상에서, 본 발명은 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 제공하고, 트랜스멤브레인 α-헬릭스 5 는 SEQ ID NO: 4 를 포함한다. 또 다른 양상에서, 본 발명은 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 제공하고, 트랜스멤브레인 α-헬릭스 6 은 SEQ ID NO: 5 를 포함한다. 또 다른 양상에서, 본 발명은 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 제공하고, 트랜스멤브레인 α-헬릭스 6 및 트랜스멤브레인 α-헬릭스 7 사이의 서열은 SEQ ID NO: 6 을 포함한다. 또 다른 양상에서, 본 발명은 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 제공하고, 트랜스멤브레인 α-헬릭스 7 은 SEQ ID NO: 7 을 포함한다. 또 다른 양상에서, 본 발명은 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 제공하고, 트랜스멤브레인 α-헬릭스 10 및 트랜스멤브레인 α-헬릭스 11 및 이들 사이의 서열은 SEQ ID NO: 8 을 포함한다.
상기한 양상의 각각은 어떠한 수의 조합(combinations)으로 결합될 수 있다. 이러한 양상에 따른 폴리뉴클레오티드는 SEQ ID NOs: 1-8 중 어떠한 것의 1, 2, 3, 4, 5, 6 또는 7 을 포함하는 폴리펩티드를 코드할 수 있거나, 폴리펩티드는 SEQ ID NOs: 1-8 의 모두를 포함할 수 있다. 예를 들어, 폴리뉴클레오티드는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩할 수 있고, 상기 트랜스멤브레인 α-헬릭스 1 은 SEQ ID NO: 1 을 포함하고, 트랜스멤브레인 α-헬릭스 2 와 트랜스멤브레인 α-헬릭스 3 을 연결하는 루프는 SEQ ID NO: 3 을 포함하고, 트랜스멤브레인 α-헬릭스 7 은 SEQ ID NO:7 을 포함한다. 또는 다른 예에서, 폴리뉴클레오티드는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩할 수 있고, 상기 트랜스멤브레인 α-헬릭스 2 는 SEQ ID NO: 2 를 포함하고, 상기 트랜스멤브레인 α-헬릭스 3 은 SEQ ID NO: 3 을 포함하고, 상기 트랜스멤브레인 α-헬릭스 6 은 SEQ ID NO: 5 를 포함하고, 트랜스멤브레인 α-헬릭스 10 과 트랜스멤브레인 α-헬릭스 11 및 이들 사이의 서열은 SEQ ID NO: 8 을 포함한다.
상기한 양상의 특정 실시형태에서, 상기 폴리펩티드는 NCU00801 또는 NCU08114 와 적어도 29 %, 적어도 30 %, 적어도 35 %, 적어도 40 %, 적어도 45 %, 적어도 50 %, 적어도 55 %, 적어도 60 %, 적어도 65 %, 적어도 70 %, 적어도 75 %, 적어도 80 %, 적어도 85 %, 적어도 90 %, 적어도 95 %, 적어도 99 %, 적어도 100 % 의 의 아미노산 동일성을 갖는다.
본원에 추가로 기재된 바와 같이, NCU08221 및 STL12/XUT6 은 자일로스를 수송하는 폴리펩티드를 코드하는 것으로 발견되었다. XUT1 은 아라비노스를 수송하는 폴리펩티드를 코드하는 것으로 발견되었다. NCU06138 은 아라비노스 또는 글루코스를 수송하는 폴리펩티드를 코드하는 것으로 발견되었다. SUT2, SUT3, 및 XUT3 은 자일로스 또는 글루코스를 수송하는 폴리펩티드를 코드하는 것으로 발견되었다. NCU04963 은 자일로스, 아라비노스 또는 글루코스를 수송하는 폴리펩티드를 코드하는 것으로 발견되었다. 바람직한 실시형태에서, 본 발명의 폴리뉴클레오티드는 NCU08221 또는 STL12/XUT6 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리펩티드는 자일로스를 수송한다. 다른 바람직한 실시형태에서, 본 발명의 폴리뉴클레오티드는 XUT1 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리펩티드는 아라비노스를 수송한다. 다른 바람직한 실시형태에서, 본 발명의 폴리뉴클레오티드는 NCU06138 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리펩티드는 아라비노스 또는 글루코스를 수송한다. 다른 바람직한 실시형태에서, 본 발명의 폴리뉴클레오티드는 SUT2, SUT3 또는 XUT3 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리펩티드는 자일로스 또는 글루코스를 수송한다. 다른 바람직한 실시형태에서, 본 발명의 폴리뉴클레오티드는 NCU04963 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리펩티드는 자일로스, 아라비노스 또는 글루코스를 수송한다.
NCU07705 에 의해 코드되는 폴리펩티드를 코드하는 본 발명의 폴리뉴클레오티드는, 분류되지 않는 단백질(unclassified protein)을 코드하기 위해 FunCat (Ruepp, 2004; webpage broad.mit.edu/annotation/genome/neurospora/Home.html)에 의해 예측되었다. 그러나, NCU07705 에 의해 코드되는 폴리펩티드의 BLAST 분석은, 전사인자(transcription factors)를 포함하는 많은 C6 징크 핑거 도메인(zinc finger domain)과 높은 유사성을 갖는 것으로 나타났다[도 1 참조 ; 모범적인 상동성(exemplary homologs)의 목록은 관련된 U.S. Appl. No. 61/271,833 의 도 23 에서 발견될 수 있다]. 본 발명의 폴리뉴클레오티드는, NCU07705 에 의해 코드되는 폴리펩티드의 이러한 상동체(homologs) 또는 본 분야에서 공지된 어떠한 방법으로 확인된 그 밖의 상동체를 코드하는 폴리뉴클레오티드를 포함한다.
본 발명의 또 다른 양상에서, 본 발명의 폴리뉴클레오티드는 NCU05137 에 의해 코드되는 폴리펩티드를 코드하는 이러한 폴리뉴클레오티드를 포함한다. FunCat 은 분류되지 않는 단백질(unclassified protein)인 NCU05137 에 의해 코드되는 폴리펩티드를 분류한다. 그러나, NCU05137 은 많은 실모양의 자낭기 균류(filamentous ascomycete fungi)의 게놈에 매우 잘 보존되었다(도 2 참조). 본 발명의 폴리뉴클레오티드는, NCU05137 에 의해 코드되는 폴리펩티드의 이러한 상동체 또는 본 분야에서 공지된 모든 방법으로 확인된 그 밖의 상동체를 코드하는 폴리뉴클레오티드를 포함한다.
본 발명의 또 다른 양상에서, 본 발명의 폴리뉴클레오티드는, NCU01517, NCU09133 또는 NCU10040 에 의해 코드되는 폴리펩티드를 코드하는 이러한 폴리뉴클레오티드를 포함한다. FunCat 은 글루코아밀라아제 전구체(glucoamylase precursor)가 될 예정인 NCU01517 에 의해 코드되는 폴리펩티드를 분류한다. FunCat 은 분류되지 않은 단백질이 될 예정인 NCU09133 및 NCU10040 에 의해 코드되는 폴리펩티드를 분류한다. 본 발명의 폴리뉴클레오티드는, NCU01517, NCU09133 또는 NCU10040 에 의해 코드되는 폴리펩티드의 이러한 상동체 또는 본 분야에서 공지된 어떠한 방법으로 확인된 그 밖의 상동체를 코드하는 폴리뉴클레오티드를 포함한다.
이러한 폴리펩티드의 예측된 기능은 폴리뉴클레오티드 및 이의 코드된 단백질의 기능적인 분석(functional analyses)을 실시하여 확인할 수 있다. 이러한 분석은, 예를 들어 폴리뉴클레오티드의 결실(deletions)을 포함하는 스트레인의 표현형 분석(phenotypic analysis), 유전적인 상보성 실험(genetic complementation experiments), 폴리뉴클레오티드의 야생형 복제(wild-type copy)가 발현된 스트레인의 표현형 분석, 폴리펩티드의 재조합 형태의 발현 및 정제, 및 재조합 폴리펩티드의 생화학적인 특성 및 활성도의 차후의 특성을 포함할 수 있다.
본 발명의 폴리뉴클레오티드의 서열은, 예를 들어 직접적인 화학적 합성(direct chemical synthesis) 또는 클로닝(cloning)을 포함하는, 본 분야의 숙련자에게 공지된 적절한 방법으로 제조할 수 있다. 직접적인 화학적인 합성에 대해서, 핵산의 중합체의 형성은, 성장하는 뉴클레오티드 사슬(growing nucleotide chain)의 말단 5'-히드록실기에 3'-블록된(blocked) 및 5'-블록된 뉴클레오티드 단량체(nucleotide monomers)의 순차적인 첨가(sequential addition)를 일반적으로 포함하고, 포스포트리에스테르(phosphotriester), 포스포르아미디트(phosphoramidite)와 같은 일반적인 인 유도체(phosphorus derivative)인, 상기 각각의 첨가는, 첨가된 단량체의 3'-위치에서 성장하는 사슬의 말단 5'-히드록실기의 핵친화성 공격(nucleophilic attack)에 의해 영향받는다. 이러한 방법론(methodology)은 본 분야에서 일상적인 기술로 알려져 있고, 적절한 문맥 및 문헌에 기재되어 있다[예를 들어, in Matteucci et al ., (1980) Tetrahedron Lett 21:719-722; U.S. Pat. Nos. 4,500,707 ; 5,436,327 ; 및 5,700,637]. 게다가, 목적하는 서열은, 적절한 제한 효소를 사용한 DNA 분리(splitting), 겔 전기영동을 사용한 단편의 분리, 및 그 후에 중합효소 연쇄 반응(polymerase chain reactions, PCR; 예를 들어, U.S. Pat. No. 4,683,195)의 이용과 같은 본 분야의 공지된 기술을 통한 겔(gel)로부터의 목적하는 핵산 서열의 회복(recovering)에 의한 천연 자원(natural sources)으로부터 분리될 수 있다.
본 발명의 각각의 뉴클레오티드는 발현 벡터(expression vector) 내로 포함될 수 있다. "발현 벡터" 또는 "벡터"는 숙주 세포에 형질도입되거나(transduces), 형질전환되거나(transforms) 또는 침투한(infect) 화합물(compound) 및/또는 조성물(composition)을 나타낸 것이고, 이에 의해서 세포에 천연 이외의 또는 세포에 천연이 아닌 방식으로 핵산 및/또는 단백질이 발현되는 세포가 야기된다. "발현 벡터" 는 숙주 세포에 의해 발현될 핵산의 서열(보통 RNA 또는 DNA)을 포함한다. 선택적으로, 벡터의 발현은, 바이러스, 리포솜(liposome), 단백질 코팅(protein coating) 또는 그 밖에 유사한 것과 같은 숙주 세포 내로의 핵산의 유입을 달성하는데 이를 돕는 물질을 또한 포함한다. 본 발명에 사용에 예상된 발현 벡터는, 바람직하거나 또는 요구되는 요소와 함께 핵산 서열을 삽입할 수 있는 이러한 것들을 포함한다. 추가로, 발현 벡터는, 숙주 세포 내로 이동될 수 있고 그 안에 복제될 수 있는 것이어야 한다. 바람직한 발현 벡터는 플라스미드, 특히 핵산 서열의 전사에 바람직하거나 요구되는 사용할 준비가 갖춰진 요소(operational elements)를 포함하고, 잘 기록된 제한효소 절단위치(restriction sites)를 갖는 이러한 것들이다. 그 밖의 발현 벡터 뿐만 아니라 이러한 플라스미드는 본 분야에 널리 공지되어 있다.
개별적인 폴리뉴클레오티드의 혼합은, 발현벡터, 예를 들어 플라스미드에서의 특정한 부위(specific sites)를 분해하기 위해서, 예를 들어 제한효소(BamRl, EcoRl, Hhal, Xhol, Xmal, 및 등과 같은)의 사용을 포함하는 공지된 방법을 통해서 실시할 수 있다. 제한효소(restriction enzyme)는 분리된 발현벡터의 말단에 대한 서열 상보성을 갖는 말단을 갖기 위해 합성하거나 또는 이를 갖는 폴리뉴클레오티드에 어닐링(annealed)될 수 있는 단일 가닥 말단(single stranded ends)을 생산한다. 어닐링(Annealing)은 적절한 효소, 예를 들어 DNA 리가아제(ligase)를 사용하여 실시하였다. 본 분야에서의 보통의 기술의 이러한 것들에 의해 예측될 수 있는 바와 같이, 발현 벡터 및 목적하는 폴리뉴클레오티드 둘 다는 동일한 제한 효소로 보통 분리되었고, 이에 의해서 발현 벡터의 말단 및 폴리뉴클레오티드의 말단은 서로 상보적임을 확인할 수 있다. 게다가, DNA 링커는 발현 벡터 내로 핵산 서열의 연결(linking)을 가능하게 하는데 사용될 수 있다.
개별적인 폴리뉴클레오티드는 본 분야에서의 기술 분야에서 공지된 방법을 사용하여 또한 결합할 수 있다(예를 들어, U.S. Pat. No. 4,683,195).
예를 들어, 각각의 목적하는 폴리뉴클레오티드는 분리된 PCR(separate PCR)에서 초기에 생성될 수 있다. 그 후에, 특정한 프라이머는, PCR 산물의 말단은 상보적인 서열(complementary sequences)을 포함하는 것과 같이 설계되었다(design). PCR 산물이 혼합되고, 변성되었고(denatured), 다시아닐되었을 때(reannealed), 이들의 3' 말단에 매칭 서열(matching sequences)을 갖는 가닥이 겹쳐지고(overlap) 서로에 대한 프라이머로서 작용할 수 있다. DNA 중합효소에 의한 이러한 오버랩(overlap)의 확대는, 원본 서열이 서로 "이어지는(spliced)" 분자를 생성한다. 이러한 방식에서, 개별적인 폴리뉴클레오티드의 시리즈(series)는 서로 "이어질 수 있고", 동시에 숙주 세포 내로 형질도입되었다(transduced). 따라서, 각각의 많은 수의 폴리뉴클레오티드의 발현에 영향을 미친다.
각각의 폴리뉴클레오티드 또는 "이어진(spliced)" 폴리뉴클레오티드는 발현벡터 내로 포함된다. 본 발명은 폴리뉴클레오티드가 발현 벡터 내로 포함되는 과정에 대해서 제한하지 않는다. 본 분야에서의 기술 분야는 발현 벡터 내로 폴리뉴클레오티드를 포함하는 필수단계와 익숙하다. 일반적인 발현 벡터는, 리보솜 결합 부위(ribosome binding site)와 함께, 하나 또는 그 이상의 조절영역(regulatory regions)에 의해 앞서는(preceded) 목적하는 폴리뉴클레오티드, 예를 들어 E. coli.에서 길이에서 3-9 뉴클레오티드이고, 개시코돈(initiation codon)의 3-11 뉴클레오티드의 업스트림(upstream)에 위치한 뉴클레오티드 서열을를 포함한다. Shine and Dalgarno (1975) Nature 254(5495):34-38 and Steitz (1979) Biological Regulation and Development (ed. Goldberger, R. F.), 1:349-399 (Plenum, New York)을 참조하라.
본원에서 사용된 바와 같은 용어 "작동가능하게 연결된(operably linked)"은, 조절 서열(control sequence)이 폴리펩티드 발현을 지시하는 것과 같이, 조절 서열이 DNA 서열 또는 폴리뉴클레오티드의 코딩 서열(coding sequence)에 관하여 적절한 위치에 놓인, 배열(configuration)을 나타낸 것이다.
조절 영역(Regulatory regions)은 예를 들어, 프로모터 및 작동유전자(operator)를 포함하는 이러한 영역을 포함한다. 프로모터는 목적하는 폴리뉴클레오티드에 작동가능하게 연결되었고, 이에 의해서 RNA 폴리메라제 효소(polymerase enzyme)를 통해 폴리뉴클레오티드의 전사(transcription)를 개시할 수 있다. 작동유전자는 프로모터에 인접한 핵산 서열이고, 이는 억제 단백질(repressor protein)이 결합할 수 있는 단백질-결합 도메인을 포함한다. 억제 단백질의 부재에서, 전사는 프로모터를 통해서 개시한다. 나타났을 때, 억제 단백질은 작동유전자에 결합하는 작동유전자의 단백질-결합 도메인에 특이적이고, 이에 의해 전사는 저해된다. 이러한 방식에서, 전사의 조절은, 사용된 특정한 조절 영역 및 이에 대응하는 억제 단백질(repressor protein)의 존재 또는 부재를 기반으로, 성취되었다. 예는 락토오스 프로모터[Lad 억제 단백질은 락토오스와 결합하였을 때 입체구조(conformation)가 변하고 이에 의해서 작동유전자에 결합으로부터 Lad 억제 단백질을 막을 수 있다] 및 트립토판 프로모터(트립토판과 복합되었을 때, TrpR 억제 단백질은 작동유전자에 결합하는 입체구조를 갖는다 ; 트립토판의 부재에서 TrpR 억제 단백질은 작동유전자에 결합하지 않는 입제구조를 갖는다)를 포함한다. 또 다른 예는 tac 프로모터이다(de Boer et al ., (1983) Proc Natl Acad Sci USA 80(1):21-25 를 참조). 본 분야에서의 기술 분야에서 인정된 바와 같이, 이러한 및 그 밖의 발현 벡터는 본 발명에서 사용될 수 있고, 본 발명은 이러한 측면으로 한정하지 않는다.
어떠한 적절한 발현 벡터는 목적하는 서열을 포함하는데 사용될 수 있을지라도, 쉽게 입수가능한 박현 벡터는 pSClOl, pBR322, pBBRlMCS-3, pUR, pEX, pMRlOO, pCR4, pBAD24, pUC19 와 같은 플라스미드 ; Ml 3 파지(Ml 3) 및 X 파지와 같은 박테리오파아지 를 포함하지만 이로 제한되지 않는다. 물론, 이러한 발현 벡터는 특정한 숙주 세포에 대해서만 적절할 수 있다. 그러나 본 분야의 기술분야 중 하나는, 어떠한 특정 발현 벡터가 어떠한 주어진 숙주 세포에 적절한지 여부를 일상적인 실험을 통해 쉽게 결정할 수 있다. 예를 들어, 발현 벡터를 숙주 세포 내로 도입할 수 있고, 이는 벡터에 포함된 서열의 발현 및 실행가능성(viability)에 대해서 모니터하였다. 게다가, 참조문헌은 관련된 문맥 및 문헌을 포함하고, 이는 어떠한 특정한 숙주 세포에 대한 발현 벡터 및 이들의 적합성(suitability)을 기재하였다.
본 발명의 숙주 세포
본원의 본 발명은 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포에 관한 것이고, 상기 폴리펩티드는 세포 내로 셀로덱스트린 또는 오탄당을 수송한다. 본원에 기재된 추가적인 발명은 숙주 세포 내로 셀로덱스트린 수송을 증가시키는 방법, 셀로덱스트린을 포함하는 배지에서 숙주 세포의 성장을 증가시키는 방법, 셀룰로스-유도 및 헤미셀룰로스-유도 당을 공동-발효시키는 방법 및 세포 내로 셀로덱스트린을 수송하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하여 탄화수소 또는 탄화수소 유도체를 생성하는 방법에 관한 것이다. 본원에 기재된 추가적인 발명은 숙주 세포 내로 오탄당의 수송을 증가시키는 방법, 오탄당 당을 포함하는 배지에 숙주 세포의 성장을 증가시키는 방법, 세포 내로 오탄당을 수송하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하여 탄화수소 또는 탄화수소 유도체를 생성하는 방법에 관한 것이다.
"숙주 세포" 및 "숙주 미생물(host microorganism)"은, 재조합 DNA 또는 RNA 의 삽입을 통해 형질전환시킬 수 있는 살아있는 생물학적 세포(living biological cell)에 관한 것에 대해서 본원에서 교환할 수 있게(interchangeably) 사용되었다. 이러한 재조힙 DNA 또는 RNA 는 발현 벡터 일 수 있다. 따라서, 본원에 기재된 바와 같은 숙주 유기체 또는 세포는 원핵 유기체(prokaryotic organism)[예를 들어, 세균계(kingdom Eubacteria)의 유기체] 또는 진핵 세포(eukaryotic cell)이다. 본 분야의 기술 분야에 의해 예측될 수 있는 바와 같이, 원핵세포는 막-결합 핵(membrane-bound nucleus)이 결핍되어 있는 반면에, 진핵세포는 막-결합 핵을 갖는다.
어떠한 원핵 또는 진핵 숙주 세포는, 핵산의 서열로 형질전환된(transformed) 후에 실행 가능하게 남아있기만 하면 본 발명에서 사용될 수 있다. 바람직하게, 숙주 세포는 필수적인 핵산 서열의 형질도입(transduction), 그 다음의 단백질의 발현(예를 들어 운반체), 또는 결과적으로 생성된 중간물질(intermediates)에 의해 불리하게 영향을 받지 않는다. 적절한 진핵 세포는 균류(fungal), 식물, 곤충 또는 포유류의 세포를 포함하지만 이로 제한되지 않는다.
바람직한 실시형태에서, 숙주 세포는 균류 스프레인(fungal strain)이다. 본원에 사용된 바와 같은 "균류(fungi)"은 문(phyla) 자낭균류(Ascomycota), 담자균류(Basidiomycota), 호상균류(Chytridiomycota) 및 접합균류(Zygomycota)(as defined by Hawksworth et al ., In, Ainsworth and Bisby's Dictionary of The Fungi, 8th edition, 1995, CAB International, University Press, Cambridge, UK), 뿐만 아니라 난균강(Oomycota)(Hawksworth et al ., 1995, supra, page 171 에 인용된) 및 모든 불완전균류(mitosporic fungi)(Hawksworth et al ., 1995, supra)를 포함한다
특정 실시형태에서, 균류 숙주(fungal host)는 효소 스트레인이다. 본원에서 사용된 바와 같은 "효모" 은 자낭포자형성효모(ascosporogenous yeast)[자낭균 효모(Endomycetales)], 담자포자효모(basidiosporogenous yeast), 및 불완전균류(Fungi Imperfecti)[불완전효모강(Blastomycetes)]에 속하는 효모를 포함한다. 효모의 분류는 앞으로도 변화될 수 있기 때문에, 본 발명의 목적에서, 효모는 Biology and Activities of Yeast 에 기재된 바와 같이 규정될 수 있다(Skinner, F. A., Passmore, S. M., and Davenport, R. R., eds, Soc. App. Bacteriol. Symposium Series No. 9, 1980).
보다 바람직한 실시형태에서, 효모 숙주는 칸디다( Candida ), 한제눌라( Hansenula ), 클루이베르마이세스 ( Kluyveromyces ), 피치아 ( Pichia ), 사카로미세스( Saccharomyces ), 스키조사카로미세스 ( Schizosaccharomyces ) 또는 야로이 야( Yarrowia ) 스트레인이다.
특정 실시형태에서, 효모 숙주는 사카로미세스 칼스베르겐시스( Saccharomyces carlsbergensis )(Todkar, 2010), 사카로미세스 세레비시아( Saccharomyces cerevisiae )(Duarte et al., 2009), 사카로미세스 다아스타티쿠스( Saccharomyces diastaticus ), 사카로미세스 도우글라시아(Saccharomyces douglasii), 사카로미세스 클루이베리 ( Saccharomyces kluyveri), 사 카로미세스 노 르벤시스( Saccharomyces norbensis ), 사카로미세스 모나센시스(Saccharomyces monacensis)(GB-Analysts Reports, 2008), 사카로미세스 바야누스( Saccharomyces bayanus)(Kristen Publicover, 2010), 사카로미세스 파스토리아누스( Saccharomyces pastorianus)(Nakao et al., 2007), 사카로미세스 폼베( Saccharomyces pombe)(Mousdale, 2008), 또는 사카로미세스 오비포르미스( Saccharomyces oviformis) 스트레인이다. 그 밖의 바람직한 실시형태에서, 효모 숙주는 클루이베르 마이세스 락티스 ( Kluyveromyces lactis )(O.W. Merten, 2001), 클루이베르마이세스 프라길리스( Kluyveromyces fragilis )(Pestal et al., 2006; Siso, 1996), 클루이베르마이세스 마르시암우스( Kluyveromyces marxiamus )(K. Kourkoutas et al., 2008), 피치아 스티피티스( Pichia stipitis)(Almeida et al., 2008), 칸디다 쉬하테 ( Candida shehatae )(Ayhan Demirbas, 2003) 또는 칸디다 트로피칼리스( Candida tropicalis )(Jamai et al., 2006)이다. 다른 실시형태에서, 효모 숙주는 야로이야 리폴리티카( Yarrowia lipolytica )(Biryukova E.N., 2009), 브레타노미세스 쿠스테르시( Brettanomyces custersii )(Spindler D.D. et al., 1992) 또는 지고삭카로마이 세스 룩시( Zygosaccharomyces roux )(Chaabane et al., 2006)일 수 있다.
또 다른 특정한 실시형태에서, 균류 숙주는 곰팡이 스트레인(filamentous fungal strain)이다. "곰팡이(Filamentous fungi)"는 세분(subdivision) 진균문(Eumycota) 및 난균강(Oomycota)의 모든 실 모양의 형태(all filamentous forms)를 포함한다(Hawksworth et al ., 1995, supra 에 의해 정의된 바와 같은). 곰팡이는, 키틴, 셀룰로스, 글루칸, 키토산(chitosan), 만난 및 그 밖의 복합 다당류(complex polysaccharides)로 이루어진 균사체의 웰(mycelial wall)로 일반적으로 특징지어진다. 영양 성장(Vegetative growth)은 균사의 연장(hyphal elongation)에 의하고, 탄소 이화작용(carbon catabolism)은 절대 호기성(obligately aerobic)이다. 그에 반하여, 사카 로미세스 세레비시아와 같은 효소에 의한 영양 성장은 단세포체(unicellular thallus)의 출아(budding)에 의한 것이고, 탄소 이화작용은 발효에 의한 것이다(fermentative).
바람직한 실시형태에서, 곰팡이 숙주는, 아크레모늄 ( Acremonium ), 아스페르길루스( Aspergillus ), 푸사리움 ( Fusarium ), 후미콜라( Humicola ), 무코르 ( Mucor ), 마이셀로프오라( Myceliophthora ), 네우로스포라 ( Neurospora ), 페니실리움 ( Penicillium ), 스시탈리디움( Scytalidium ), 티엘라비아 ( Thielavia ), 톨리포클라 디움( Tolypocladium ) 또는 트리코데리마 ( Trichoderma ) 스트레인이지만, 이로 한정되지 않는다.
특정 실시형태에서, 곰팡이 숙주는, 아스페르길루스 아와모리( Aspergillus awamori), 아스페르길루스 포에티두스 ( Aspergillus foetidus ), 아스페르길루스 자포니쿠스( Aspergillus japonicus ), 아스페르길루스 니둘란스 ( Aspergillus nidulans ), 아스페르길루스 니가 ( Aspergillus niger ), 또는 아스페르길루스 오리재( Aspergillus oryzae ) 스트레인이다. 다른 실시형태에서, 곰팡이 숙주는 푸사리움 박트리디오이데스 ( Fusarium bactridioides ), 푸사리움 세렐리스( Fusarium cerealis ), 푸사리움 크룩웰렌스 ( Fusarium crookwellense ), 푸사리움 쿨모룸( Fusarium culmorum ), 푸사리움 그래미니아룸 ( Fusarium graminearum ), 푸사 리움 그래미눔 ( Fusarium graminum ), 푸사리움 헤테로스포룸( Fusarium heterosporum), 푸사리움 네군디 ( Fusarium negundi ), 푸사리움 옥시스포룸( Fusarium oxysporum ), 푸사리움 레티쿨라툼 ( Fusarium reticulatum ), 푸사리움 로세움( Fusarium roseum ), 푸사리움 삼부시눔 ( Fusarium sambucinum ), 푸사리움 사로크로움( Fusarium sarcochroum ), 푸사리움 스포로트리치오이데스( Fusarium sporotrichioides), 푸사리움 술푸레움 ( Fusarium sulphureum ), 푸사리움 토룰로숨( Fusarium torulosum ), 푸사리움 트리초테시오이데스( Fusarium trichothecioides), 또는 푸사리움 베네나툼 ( Fusarium venenatum ) 스트레인이다. 또 다른 바람직한 실시형태에서, 곰팡이 숙주는 후미콜라 인솔렌스( Humicola insolens), 후미콜라 라누기노사 ( Humicola lanuginosa ), 무코르 미에헤이( Mucor miehei), 마이셀로프토라 서모필라 ( Myceliophthora thermophila ), 네우로스포라 크라사( Neurospora crassa ), 페니실리움 푸르푸로게눔 ( Penicillium purpurogenum ), 스시탈리디움 서모필리움 ( Scytalidium thermophilum ), 스포로트리쿰 서모필( Sporotrichum thermophile )( Topakas et al ., 2003), 또는 티엘라비아 테레스트 리스( Thielavia terrestris ) 스트레인이다. 추가적인 실시형태에서, 곰팡이 숙주는 트리코델마 하리지아눔 ( Trichoderma harzianum ), 트리코데르마 코닌기( Trichoderma koningii), 트리코데르마 롱기브라치아툼 ( Trichoderma longibrachiatum), 트리코데리마 레세이 ( Trichoderma reesei ) 또는 트리코데르마 비리드( Trichoderma viride ) 스트레인이다.
그 밖의 바람직한 실시형태에서, 숙주 세포는 원핵(prokaryotic)이고, 특정 실시형태에서, 원핵생물은 E. coli(Dien, B.S. et al., 2003; Yomano, L. P. et al., 1998; Moniruzzaman et al., 1996), 고초균( Bacillus subtilis )(Susana Romero et al., 2007), 자이모모나스 모빌리스( Zymomonas mobilis )(B. S. Dien et al, 2003; Weuster Botz, 1993; Alterthum and Ingram, 1989), 클로스트리 듐( Clostridium sp .)(Zeikus, 1980; Lynd et al., 2002; Demain et al., 2005), 클로스트리듐 파이토퍼멘탄스 (Clostridium phytofermentans)(Leschine S., 2010), 클로스트리듐 서모셀룸 ( Clostridium thermocellum )(Lynd et al., 2002), 클로스트리듐 베이저린키이( Clostridium beijerinckii )(Giles Clark, 2008), 클로스트리듐 아세토부틸리쿰( Clostridium acetobutylicum )[ 무렐라 서모아세티카( Moorella thermoacetica)](Huang W.C. et al., 2004; Dominik et al., 2007), 서모아네로박 테리움 사카로락티쿰 ( Thermoanaerobacterium saccharolyticum )(Marietta Smith, 2009) 또는 켈브시엘라 옥시토카( Klebsiella oxytoca )(Dien, B.S. et al., 2003; Zhou et al., 2001; Brooks and Ingram, 1995)이다. 또 다른 실시형태에서, 원핵 숙주 세포는 카르복시도셀라 ( Carboxydocella sp .)(Dominik et al., 2007), 코리네 박테리움 글루타미쿰 ( Corynebacterium glutamicum )(Masayuki Inui, et al., 2004), 엑테로박테리아세아에( Enterobacteriaceae )(Ingram et al., 1995), 어르위니아 크리산테미( Erwinia chrysanthemi )(Zhou and Ingram, 2000; Zhou et al., 2001), 락토바실리우스( Lactobacillus sp .)(McCaskey, T.A., et al., 1994), 페디오코쿠스 아시디락티시( Pediococcus acidilactici )(Zhou, S. et al., 2003), 로도프세우도모 나스 캡슐라타 ( Rhodopseudomonas capsulata )(X.Y. Shi et al., 2004), 스트렙토코크스 락토스( Streptococcus lactis )(J.C. Tang et al., 1988), 비브리오 푸르니시( Vibrio furnissii )(L.P. Wackett, 2010), 비브리오 푸르니시 M1( Vibrio furnissii Ml)(Park et al, 2001), 칼디셀룰로시루프토르 사카로락티쿠스( Caldicellulosiruptor saccharolyticus )(Z. Kadar et al., 2004), 또는 잔토모 나스 캄페스트리스( Xanthomonas campestris )(S.T. Yang et al., 1987)이다. 또 다른 실시형태에서, 숙주 세포는 시아노박테리아(cyanobacteria)이다. 박테리아 숙주 세포의 추가적인 예는 에세리키아 ( Escherichia ), 엔테로박터( Enterobacter ), 아조토박터( Azotobacter), 에르위니아 ( Erwinia ), 바실루스 ( Bacillus ), 프세우도모나스 ( Pseudomonas ), 클렙시엘라( Klebsiella ), 프로테우스( Proteus ), 살모넬라( Salmonella ), 세라티아( Serratia ), 쉬겔라( Shigella ), 리조비아 ( Rhizobia ), 비트레오스실라 ( Vitreoscilla ), 시네초코커스( Synechococcus ), 시네초시스티 스( Synechocystis ), 및 파라코커스 ( Paracoccus ) 탁솔노미칼 클래스(taxonomical classes)를 포함하지만 이로 제한되지 않는다.
본 발명의 특히 바람직한 실시형태에서, 상기 숙주 세포는 사카로마이시스( Saccharomyces sp .), 사카로미세스 세레비시아 ( Saccharomyces cerevisiae ), 사카로미세스 모나센시스( Saccharomyces monacensis ), 사카로미세스 바야누스 ( Saccharomyces bayanus ), 사카로미세스 파스토리아누스 ( Saccharomyces pastorianus ), 사카로미세스 칼스베르겐시스( Saccharomyces carlsbergensis ), 사카로미세스 폼베 ( Saccharomyces pombe ), 클루이베르마이세스( Kluyveromyces sp .), 클루이베르마이세스 마르시암우스 ( Kluyveromyces marxiamus ), 클루이베르마이세스 락티스 ( Kluyveromyces lactis ), 클루이베르마이세스 프라길리스( Kluyveromyces fragilis), 피치아 스티피티스 ( Pichia stipitis ), 스포로트리쿰 서머파일( Sporotrichum thermophile), 칸디다 쉬하테 ( Candida shehatae ), 칸디다 트로피칼리스 ( Candida tropicalis ), 네우로스포라 크라사 ( Neurospora crassa ), 자이모나스 모빌리스( Zymomonas mobilis ), 클로스트리듐( Clostridium sp .), 클로스트리듐 파이토퍼멘탄스 ( Clostridium phytofermentans ), 클로스트리듐 써모셀럼( Clostridium thermocellum ), 클로스트리듐 베이저린키이( Clostridium beijerinckii), 클로스트리듐 아세토부틸리쿰 ( Clostridium acetobutylicum ), 무렐 라 서모아세티카 ( Moorella thermoacetica ), 대장균( Escherichia coli ), 크레브시엘라 옥시토카 ( Klebsielle oxytoca ), 서모안아에로박테리움 사카로락티 쿰( Thermoanaerobacterium saccharolyticum ) 및 고초균( Bacillus subtilis )이다. 사카로마이시스는 산업적인(Industrial) 사카로마이시스 스트레인을 포함할 수 있다. Argueso et al. 은 바이오에탄올 생산(bioethanol production)에 중요한 특정한 유전자 다형성(specific gene polymorphisms) 뿐만 아니라 바이오에탄올 생산에 일반적으로 사용되는 산업적인 사카로마이시스 스트레인의 게놈 구조를 논의하였다(Genome Research, 19: 2258-2270, 2009).
본 발명의 숙주 세포는, 재조합 핵산을 숙주 세포 내로 도입하는, 유전적으로 형질전환될 수 있고, 유전적으로 형질전환된 숙주 세포는 자연적으로 발생하지 않는다. 적절한 숙주 세포는 상이한 기능에 대해서 하나 또는 그 이상의 단백질을 코딩하는 하나 또는 그 이상의 핵산 구조(nucleic acid constructs)가 발현될 수 있는 것이다.
본원에서 사용된 바와 같은 "재조합 핵산" 또는 "이형 핵산(heterologous nucleic acid)" 또는 "재조합 폴리뉴클레오티드"는 핵산 중합체에 관한 것이고, 하기의 적어도 하나는 사실(true)이다 : (a) 핵산의 서열이 주어진 숙주 세포와 이질적이다(foreign)(즉, 숙주 세포에서 천연적으로 발견되지 않는다) ; (b) 서열이 주어진 숙주 세포에서 천연적으로 발견되지만, 천연적이지 않는 양(unnatural amount)(예를 들어, 예상했던 양보다 많이)으로 발견되었다 ; 또는 (c) 핵산의 서열이, 천연적으로(in nature) 서로에 대해 동일한 관계(same relationship)로 발견되지 않는 둘 또는 그 이상의 서브서열(subsequences)을 포함한다. 예를 들어, 경우 (c) 에 관하여, 재조합 핵산 서열은, 신규한 기능적인 핵산을 생산하기 위해 배열된 관련되지 않은 유전자(unrelated genes)로부터 둘 또는 그 이상의 서열을 갖는다. 명확하게, 본 발명은 숙주 세포 내로 발현 벡터의 도입(introduction)을 기재하였고, 상기 발현 벡터는, 숙주 세포에서 일반적으로 발견되지 않는 단백질을 코딩하는 핵산 서열을 포함하거나, 또는 숙주 세포에서 일반적으로 발견되지만 상이한 조절 서열(regulatory sequences)의 조절 하에 있는 단백질을 코딩하는 핵산을 포함한다. 숙주 세포의 게놈과 관련하여, 단백질을 코드하는 상기 핵산 서열은 재조합되었다(recombinant).
몇몇 실시형태에서, 상기 숙주 세포는 본 발명의 폴리뉴클레오티드에 의해 코드되는 모든 단백질을 천연적으로 생산한다. 목적하는 단백질을 코딩하는 유전자는 숙주 세포에 대해서 이종 기원(heterologous)일 수 있거나, 또는 이러한 유전자는 숙주 세포에 대해서 내인적(endogenous)일 수 있지만, 이종 기원의 프로모터(heterologous promoters)에 작동가능하게 연결되고/연결되거나 숙주 세포에서 유전자의 보다 높은 발현을 야기하는 영역을 조절한다. 다른 실시형태에서, 숙주 세포는 목적하는 단백질을 천연적으로 생산하지 않고, 이러한 분자를 생산하기 위해 필요한 하나 또는 그 이상의 유전자를 발현시킬 수 있는 이종 기원 핵산 구조물(heterologous nucleic acid constructs)을 포함한다.
핵산 분자 또는 폴리펩티드 및 특정한 세포 또는 미생물과 관련하여 본원에서 사용된 바와 같은 "내인성(Endogenous)" 은 재조합 유전자 조작 기술(recombinant engineering techniques)을 사용한 세포 내로 도입되지 않고, 세포 내에 있는 핵산 서열 또는 펩티드에 관한 것이다 ; 예를 들어 세포를 천연으로부터 본래 분리하였을 때, 세포에 존재하는 유전자.
"유전적으로 조작된(Genetically engineered)" 또는 "유전적으로 변형된(genetically modified)"은, 높은 수준(elevated levels), 낮은 수준(lowered levels)으로 또는 돌연변이된 형태(mutated form)로 단백질을 발현시키는 원핵 또는 진핵 숙주 세포를 생산하는데 사용된 모든 재조합 DNA 또는 RNA 방법에 관한 것이다. 다시 말해서, 숙주 세포를 재조합 폴리뉴클레오티드 분자로 감염시기키거나(transfected), 형질전환시키거나(transformed), 또는 형질 도입시킨 것으로(transduced), 이것 때문에 목적하는 단백질의 발현을 변경시키는 세포가 발생하여 변경되었다. 유전적으로 조작된 숙주 세포에 대한 방법 및 벡터는 본 분야에서 널리 공지되어 있다 ; 예를 들어 다양한 기술은 Molecular Biology, Ausubel et al., eds. (Wiley & Sons, New York, 1988, and quarterly updates)에서의 현재의 프로토콜에 기재되어 있다. 유전적으로 조작된 기술(Genetically engineering techniques)은, 발현 벡터, 목표하는 상동 재조합(targeted homologous recombination) 및 유전자 활성화(gene activation)(예를 들어, U.S. Pat. No. 5,272,071 to Chappel 참조) 및 유전자 조작된 전사 인자에 의한 트랜스- 활성화(trans- activation)[예를 들어, Segal et al ., (1999) Proc Natl Acad Sci USA 96(6):2758-2763 참조]를 포함하지만 이로 제한되지 않는다.
유전자 발현 또는 기능에서 증가를 야기하는 유전적인 변형(Genetic modifications)은, 유전자의 증폭(amplification), 과잉생산(overproduction), 과잉발현(overexpression), 활성화(activation), 증대(enhancement), 첨가 또는 상향-조절(up-regulation)로 언급될 수 있다. 보다 명확하게, 본원에 기재된 효소 또는 그 밖의 단백질의 작용(action)(또는 활성도)를 증가시키는 것과 관련하여, 효소 또는 단백질의 증가된 발현 및/또는 기능성(생물학적 활성도)를 야기하는 숙주 세포의 어떠한 유전적인 변형을 일반적으로 언급하고, 단백질의 보다 높은 활성도 또는 작용(예를 들어, 특이적인 활성도 또는 생체 내 효소적 활성도), 감소된 저해 또는 단백질의 분해(degradation), 및 단백질의 과잉발현을 포함한다. 예를 들어, 유전자 복제수(gene copy number)는 증가될 수 있고, 발현 수준은 천연의 프로모터의 발현 수준보다 더 높은 발현 수준을 나타내는 프로모터의 사용에 의해 증가될 수 있거나, 또는 유전자가 단백질의 작용 또는 효소의 생물학적 활성도를 증가시키기 위해 유전적으로 조작되거나 또는 일반적인 돌연변이 생성으로 변경될 수 있다. 이러한 변형의 몇몇의 조합은 또한 가능하다.
유전자 발현, 유전자의 기능 또는 유전자 산물(즉, 유전자에 의해 코드되는 단백질)의 기능에서 감소를 야기하는 유전적인 변형(genetic modification)은, 유전자 발현의 불활성화(inactivation)(완전한 또는 부분적인), 결실, 중단(interruption), 장애(blockage), 침묵(silencing), 하향-조절(down-regulation), 감쇠(attenuation)로 언급될 수 있다. 예를 들어, 이러한 유전자에 의해 코드되는 단백질의 기능에서의 감소를 야기하는 유전자에서의 유전적인 변형은, 유전자의 완전한 결실(즉, 유전자가 존재하지 않고, 따라서 단백질이 존재하지 않는다), 단백질의 불완전하거나 또는 번역이 없는 것을 야기하는 유전자에서의 돌연변이(예를 들어, 단백질이 발현되지 않는다), 또는 단백질의 천연적인 기능을 감소시키거나 폐지하는(abolish) 유전자에서의 돌연변이(예를 들어, 효소적인 활성도 또는 작용이 없거나 감소된 단백질이 발현된다)의 결과일 수 있다. 보다 명확하게, 본원에 기재된 단백질의 작용을 감소시키는 언급은, 숙주 세포에서 모든 유전적인 변형에 대해서 일반적으로 나타내었고, 단백질의 감소된 발현 및/또는 기능성(생물학적 활성도)을 야기하고, 단백질의 감소된 활성도(예를 들어, 감소된 수송), 단백질의 증가된 저해 또는 분해 뿐만 아니라 단백질의 발현의 감소 또는 저해를 포함한다. 예를 들어, 본 발명의 단백질의 작용 또는 활성도는 단백질 생산의 저지(blocking) 또는 감소, 단백질 작용의 감소, 또는 단백질 작용의 저해에 의해 감소될 수 있다. 이러한 변형의 몇몇을 또한 조합할 수 있다. 단백질 생산의 저지 또는 감소는, 성장 배지에서 유도된 화합물의 존재를 필요로 하는 프로모터의 조절 하에서 단백질을 코딩하는 유전자를 배치하는 것을 포함할 수 있다. 유도물질(inducer)이 배지로부터 대폭 감소되는 것과 같은 조건을 설정함으로써, 단백질을 코딩하는 유전자의 발현(및 이에 따른 단백질의 합성)은 정지될 수 있다(turned off). 단백질의 작용을 저지하거나 감소시키는 것은 본원에서 참조문헌으로 인용된 U.S. Pat. No. 4,743,546 에 기재된 것과 유사한 제거 기술 접근법(excision technology approach)을 사용하는 것을 또한 포함할 수 있다. 이러한 접근법을 사용하기 위해, 흥미로운 단백질을 코딩하는 유전자는, 게놈으로부터 유전자가 특이적이고, 조절된 제거를 가능하게 하는 특이적인 유전적인 서열(specific genetic sequences) 사이에 클론되었다(cloned). 제거(Excision)는, 예를 들어 U.S. Pat. No. 4,743,546 에 기재된 배양물의 배양 온도에서의 시프트(shift), 또는 몇몇 다른 물리적이거나(physical) 또는 영양상의 신호(nutritional signal)에 의해 유도된다(prompted).
일반적으로, 본 발명에 따른, 돌연변이체(mutant) 또는 변형된 단백질(modified protein)의 주어진 특성에서의 증가 또는 감소(예를 들어, 효소 활성도, 수송 화합물의 능력)는, 동일한 또는 동등한 조건(equivalent conditions) 하에서 측정되거나 또는 확립된(established), 동일한 유기체[동일한 근원(same source) 또는 부모 서열(parent sequence)로부터]로부터 유도된 야생형(즉, 보통, 변형되지 않은) 단백질의 동일한 특성과 관련하여 행해졌다. 유사하게, 유전적으로 변형된 숙주 세포의 특성의 증가 또는 감소(예를 들어, 단백질의 발현 및/또는 생물학적 활성도, 또는 산물의 생산)는, 동일하거나 또는 동등한 조건 하에서, 동일한 종 및 바람직하게 동일한 스트레인의 야생형 숙주 세포의 동일한 특성과 관련하여 행해졌다. 이러한 조건은, 단백질의 활성도(예를 들어, 발현 또는 생물학적 활성도) 또는 숙주 세포의 그 밖의 특성을 측정하는 것 뿐만 아니라 검정 유형의 사용, 숙주 세포를 측정 하에서, 검정 또는 배양 조건(예를 들어, 배지 조성, 온도, pH 등)을 포함한다. 상기에 나타낸 바와 같이, 동등한 조건은, 유사하지만 동일하지 않고[예를 들어, 조건에서의 몇몇의 보존적인 변화(conservative changes)는 용인될 수 있다], 동일한 조건 하에서 만들어진 비교(comparison)와 비교함으로써 세포 성장 또는 효소 발현 또는 생물학적 활성도에서의 효과를 실질적으로 변화시키지 않는 조건(예를 들어, 배양 조건)이다.
바람직하게, 주어진 단백질(예를 들어, 운반체, 효소)의 활성도를 증가시키거나 감소시키는 유전적인 변형을 갖는 유전적으로 변형된 숙주 세포는, 적어도 약 5 %, 및 보다 바람직하게 적어도 약 10 %, 및 보다 바람직하게 적어도 약 15 %, 및 보다 바람직하게 적어도 약 20 %, 및 보다 바람직하게 적어도 약 25 %, 및 보다 바람직하게 적어도 약 30 %, 및 보다 바람직하게 적어도 약 35 %, 및 보다 바람직하게 적어도 약 40 %, 및 보다 바람직하게 적어도 약 45 %, 및 보다 바람직하게 적어도 약 50 %, 및 보다 바람직하게 적어도 약 55 %, 및 보다 바람직하게 적어도 약 60 %, 및 보다 바람직하게 적어도 약 65 %, 및 보다 바람직하게 적어도 약 70 %, 및 보다 바람직하게 적어도 약 75 %, 및 보다 바람직하게 적어도 약 80 %, 및 보다 바람직하게 적어도 약 85 %, 및 보다 바람직하게 적어도 약 90 %, 및 보다 바람직하게 적어도 약 95 %, 및 5 % 에서 100 % 사이의 전체 정수(예를 들어, 6 %, 7 %, 8 % 등)인 모든 퍼센트의 야생형 숙주 세포에서의 야생형 단백질의 활성도와 비교한 바와 같이, 각각 단백질의 활성도 또는 작용(예를 들어, 발현, 생산 및/또는 생물학적 활성도)에서 증가되거나 또는 감소시킨다. 분리된 야생형 핵산 분자 또는 단백질에 대해 직접적으로 분리된 변형된 핵산 분자 또는 단백질과 비교하였을 때(예를 들어, 만약 생체 내( in vivo )와 비교한 생체 외( in vitro )에서 비교한다면), 별로 다를 거 없는 것(same differences)이 바람직하다.
본 발명의 또 다른 양상에서, 주어진 단백질(예를 들어, 운반체 및 효소)의 활성도가 증가되거나 또는 감소된 유전적인 변형을 갖는 유전적으로 변형된 숙주 세포는, 적어도 약 2-배(fold), 및 보다 바람직하게 적어도 약 5-배, 및 보다 바람직하게 적어도 약 10-배, 및 보다 바람직하게 적어도 약 20-배, 및 보다 바람직하게 적어도 약 30-배, 및 보다 바람직하게 적어도 약 40-배, 및 보다 바람직하게 적어도 약 50-배, 및 보다 바람직하게 적어도 약 75-배, 및 보다 바람직하게 적어도 약 100-배, 및 보다 바람직하게 적어도 약 125-배, 및 보다 바람직하게 적어도 약 150-배, 또는 적어도 약 2-배로부터 시작하는 전체 정수 증가(integer increment)(예를 들어, 3-배, 4-배, 5-배, 6-배 등)의 야생형 숙주 세포에서의 야생형 단백질의 활성도와 비교한 바와 같이, 각각 단백질의 활성도 또는 작용(예를 들어, 발현, 생산 및/또는 생물학적 활성도)에서 증가되거나 감소되었다.
숙주 세포 성분
하나의 양상에서, 본 발명의 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하고, 상기 트랜스멤브레인 α-헬릭스 1 은 SEQ ID NO : 1 을 포함한다. 또 다른 양상에서, 본 발명의 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하고, 상기 트랜스멤브레인 α-헬릭스 2 는 SEQ ID NO : 2 를 포함한다. 또 다른 양상에서, 본 발명의 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하고, 상기 트랜스멤브레인 α-헬릭스 2 및 트랜스멤브레인 α-헬릭스 3 을 연결하는 루프는 SEQ ID NO : 3 을 포함한다. 또 다른 양상에서, 본 발명의 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하고, 상기 트랜스멤브레인 α-헬릭스 5 는 SEQ ID NO : 4 를 포함한다. 또 다른 양상에서, 본 발명의 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 6 은 SEQ ID NO : 5 를 포함한다. 또 다른 양상에서, 본 발명의 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하고, 상기 트랜스멤브레인 α-헬릭스 6 및 트랜스멤브레인 α-헬릭스 7 사이의 서열은 SEQ ID NO : 6 을 포함한다. 또 다른 양상에서, 본 발명의 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 7 은 SEQ ID NO : 7 을 포함한다. 또 다른 양상에서, 본 발명의 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 10 및 트랜스멤브레인 α-헬릭스 11 및 이들 사이의 서열은 SEQ ID NO : 8 을 포함한다.
각각의 상기한 양상은 많은 수의 조합으로 조합될 수 있다. 숙주 세포는 SEQ ID NOs: 1-8 중의 1, 2, 3, 4, 5, 6, 또는 7 을 포함하는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함할 수 있거나, 폴리펩티드는 SEQ ID NOs: 1-8 의 모두를 포함할 수 있다. 예를 들어, 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 1 은 SEQ ID NO: 1 을 포함하고, 트랜스멤브레인 α-헬릭스 2 는 SEQ ID NO : 2 를 포함하고, 트랜스멤브레인 α-헬릭스 2 및 트랜스멤브레인 α-헬릭스 3 을 연결하는 루프는 SEQ ID NO : 3 을 포함하고, 트랜스멤브레인 α-헬릭스 7 은 SEQ ID NO : 7 을 포함한다. 또는, 또 다른 예에서, 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 2 는 SEQ ID NO: 2 를 포함하고, 트랜스멤브레인 α-헬릭스 3 은 SEQ ID NO: 3 을 포함하고, 트랜스멤브레인 α-헬릭스 6 은 SEQ ID NO: 5 를 포함하고, 트랜스멤브레인 α-헬릭스 10 및 트랜스멤브레인 α-헬릭스 11 및 이들 사이의 서열은 SEQ ID NO : 8 을 포함한다.
상기한 양상의 특정 실시형태에서, 상기 폴리펩티드는 NCU00801 또는 NCU08114 와 적어도 29 %, 적어도 30 %, 적어도 35 %, 적어도 40 %, 적어도 45 %, 적어도 50 %, 적어도 55 %, 적어도 60 %, 적어도 65 %, 적어도 70 %, 적어도 75 %, 적어도 80 %, 적어도 85 %, 적어도 90 %, 적어도 95 %, 적어도 99 % 또는 적어도 100 % 의 아미노산 동일성을 갖는다.
바람직한 실시형태에서, 숙주 세포는 추가로 폴리뉴클레오티드를 포함하고, 상기 폴리뉴클레오티드는 β-글루코시다아제의 촉매 도메인(catalytic domain)을 코드한다. 본원에 사용된 바와 같이, β-글루코시다아제는 β-D-글루코시드 글루코히드롤라아제(glucohydrolase)(E.C. 3.2.1.21)을 나타내고, β-D-글루코스를 방출하는 말단 비-환원(terminal non-reducing) β-D-글루코스 잔기의 가수분해를 촉매한다. β-글루코시다아제의 촉매 도메인은, 예를 들어, Venturi et al., 2002 에 의해 기재된 기본적인 절차(basic procedure)에 따라 확인된 바와 같은 β-글루코시다아제 활성도를 갖는다. β-글루코시다아제의 촉매 도메인은 글루코스를 방출하는 β-D-글루코시드에서 말단의 비-환원 잔기의 가수분해를 촉매하는 모든 도메인이다. 바람직한 실시형태에서, β-글루코시다아제는 글리코실 가수분해효소 패밀리 1 멤버(glycosyl hydrolase family 1 member)이다. 이러한 그룹의 멤버는 모티프, [LIVMFSTC] - [LIVFYS] - [LIV] - [LIVMST] - E - N - G - [LIVMFAR] - [CSAGN] (SEQ ID NO: 14)에 의해 확인할 수 있다. 여기에서, E 는 촉매작용의 글루타메이트이다(웹페이지 expasy.org/cgi-bin/prosite-search-ac?PDOC00495). 특정 실시형태에서, β-글루코시다아제의 촉매 도메인을 코딩하는 폴리뉴클레오티드는 숙주 세포에 대해서 이종기원이다. 바람직한 실시형태에서, β-글루코시다아제의 촉매 도메인은 숙주 세포에서 세포 내에 위치한다. 바람직한 실시형태에서, β-글루코시다아제는 N. 크라사로부터 온 것이고, 특히 바람직한 실시형태에서, β-글루코시다아제는 NCU00130 이다. 특정 실시형태에서, β-글루코시다아제는 NCU00130 의 오솔로그일 수 있다. NCU00130 의 오솔로그는, T. 멜라노스포룸(T. melanosporum), CAZ82985.1; A. 오리재(A. oryzae ), BAE57671.1 ; P. 플라센타 (P. placenta ), EED81359.1; P. 크라이소스포리움 (P. chrysosporium ), BAE87009.1; 클루이베로마이세스 락티스( Kluyveromyces lactis ), CAG99696.1; 락카리아 비칼 라( Laccaria bicolor ), EDR09330; 칼라비스포라 루시타니아( Clavispora lusitaniae), EEQ37997.1; 및 피치아 스티피티스 ( Pichia stipitis ), ABN67130.1 를 포함하지만 이로 한정하지 않는다. 다른 β-글루코시다아제는 글리코실 가수분해 패밀리 3(glycosyl hydrolase family 3)으로부터 온 것을 사용할 수 있다. 이러한 β-글루코시다아제는 PROSITE 에 따른 하기의 모티프로부터 확인할 수 있다 : [LIVM](2) - [KR] - x - [EQKRD] - x(4) - G - [LIVMFTC] - [LIVT] - [LIVMF] - [ST] - D - x(2) - [SGADNIT] (SEQ ID NO: 15). 여기에서 D 는 촉매의 아스파르트산염(catalytic aspartate)이다. 일반적으로, 모든 β-글루코시다아제는 NCBI 서열 COG2723 에서 발견된 β-글루코시다아제/6-포스포-β-글루코시다아제/β-갈락토시다아제의 보존 도메인(conserved domain)을 포함한 것을 사용할 수 있다. 특정한 β-글루코시다아제로부터의 촉매 도메인은 숙주 세포에서 포함된 셀로덱스트린 운반체에 따라 선호될 수 있다.
특정 실시형태에서, 숙주 세포는 하나 또는 그 이상의 폴리뉴클레오티드를 포함하고, 상기 하나 또는 그 이상의 폴리뉴클레오티드는 오탄당 이용(pentose utilization)에서 포함되는 하나 또는 그 이상의 효소를 코드한다. 하나 또는 그 이상의 폴리뉴클레오티드는 숙주 세포에 대해서 내인성(endogenous) 또는 이종 기원(heterologous)일 수 있다. 본원에서 사용된 바와 같은 오탄당은 5 가지 탄소 원자를 갖는 모든 단당류를 나타낸다. 오탄당의 예는 자일로스, 아라비노스, 만노스, 갈락토스 및 람노오스(rhamnose)를 포함하지만 이로 한정되지 않는다. 오탄당 이용에 포함된 하나 또는 그 이상의 효소는, 예를 들어 어떠한 결합으로 L-아라비노스 이성질화효소, L-리블로인산화효소, L-리블로스-5-P 4 에피머화효소, 자일로스 이성질화효소, 자일루로키나아제, 알도스 환원효소, L-아라비니톨 4-탈수소효소, L-자일루로스 환원효소 및 자일리톨 탈수소효소를 포함할 수 있다. 이러한 효소는 오탄당 당을 천연적으로 대사작용하는 모든 유기체로부터 올 수 있다. 이러한 유기체의 예는, 예를 들어 클루이베로마이세스 ( Kluyveromyces sp.), 자이모모나스( Zymomonas sp.), E. coli , 클로스트리듐( Clostridium sp.) 및 피치아( Pichia sp.)를 포함한다.
실시예 12-15 는, 숙주 세포는 개선될 수 있거나, 보다 효율적으로 만들어질 수 있는, 오탄당 이용 경로에서의 방법을 기재한 것이다. 숙주 세포의 스트레인 백그라운드(Strain background)는 이의 오탄당 이용 경로(pentose utilization pathway)의 효율성(efficiency)에 영향을 줄 수 있다. 숙주 세포가 사카로미세스( Saccharomyces sp.)인 본 발명의 실시형태에서, 바람직한 오탄당 이용은 DA24-16(실시예 13 참조) 및 L2612(실시예 16 참조)을 포함한다. 오탄당 이용에 포함되는 효소를 코딩하는 폴리뉴클레오티드를 포함하는 그 밖의 숙주 세포는 DuPont 자이모모나스 ( Zymomonas ) 스트레인(WO 2009/058927) 및 사카로미세 스( Saccharomyces ) 스트레인(US 5,789,210)을 포함한다.
본 발명의 특정 실시형태에서, 숙주 세포는 오탄당 운반체를 코딩하는 재조합 폴리뉴클레오티드를 포함한다. 특정 실시형태에서, 오탄당 운반체는 본원에서 발견되고 기재된, NCU00821, NCU04963, NCU06138, STL12/XUT6, SUT2, SUT3, XUT1 및 XUT3(실시예 11 을 참조)을 포함하는 이러한 운반체를 포함한다. 다른 실시형태에서, 오탄당 운반체는, C. 인터메디아(C. intermedia)로부터의 Gxsl, P. 스티피티스로부터의 Autl, D. 한세니(D. hansenii)로부터의 Xylhp(Nobre et al ., 1999), K. 마르시암우스( K. marxianus)로부터의 자일로스 운반체(Stambuk et al ., 2003), 암브로시오즈마 모노스포라( Ambrosiozyma monospora )로부터의 LAT1 and LAT2(EMBL AY923868 and AY923869, respectively, R. Verho et al .), C. 아라비노페멘탄스 (C. arabinofermentans)로부터의 ART1(Fonseca et al ., 2007), K. 마르시암우스(K. marxiamus)로부터의 KmLATl(Knoshaug et al ., 2007), P. 귈리어몬디(P. guilliermondii)로부터의 PgLAT2(Knoshaug et al ., 2007), 및 P. 스티피티스(P. stipitis)로부터의 araT(Boles & Keller, 2008)을 포함할 수 있다.
본 발명의 숙주 세포를 생산하고 배양하는 방법
본원에서의 본 발명은 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포에 관한 것이고, 상기 폴리펩티드는 세포 내로 셀로덱스트린 또는 오탄당을 수송한다. 본원에 기재된 추가적인 본 발명은 숙주 세포 내로 셀로덱스트린의 수송을 증가시키는 방법, 셀로덱스트린을 포함하는 배지에서 숙주 세포의 성장을 증가시키는 방법, 셀로덱스트린-유도 및 헤미셀로덱스트린 유도 당을 공동-발효시키는 방법, 세포 내로 셀로덱스트린을 수송하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 준비하여 탄화수소 또는 탄화수소 유도체를 생성하는 방법에 관한 것이다. 본원에 기재된 추가적인 본 발명은 숙주 세포 내로 오탄당의 수송을 증가시키는 방법, 오탄당 당을 포함하는 배지에서 숙주 세포의 성장을 증가시키는 방법, 및 세포 내로의 오탄당을 수송하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 준비하여 탄화수소 또는 탄화수소 유도체를 생성하는 방법에 관한 것이다.
본 발명의 숙주 세포를 생성하고 배양하는 방법은 숙주 세포 내로 본 발명의 재조합 폴리뉴클레오티드를 포함하는 발현 벡터의 도입 또는 수송을 포함할 수 있다. 숙주 세포 내로의 발현 벡터를 수송하는 이러한 방법은 본 분야에서 기술분야에 널리 공지되어 있다. 예를 들어, 발현 벡터를 갖는 E. coli 를 변형시키는 방법은 염화칼슘 처리를 포함하고, 상기 발현 벡터는 칼슘 침전물(calcium precipitate)을 통해 도입된다. 그 밖의 염, 예를 들어 인산칼슘은 하기의 유사한 절차를 또한 사용할 수 있다. 게다가, 일렉트로포레이션(electroporation)(즉, 핵산 서열에 대한 세포의 투과성(permeability)을 증가시키기 위해 전류의 적용)을 숙주 세포를 감염시키는데(transfect) 사용될 수 있다. 또한, 핵산 서열의 미세주입법(microinjection)은 숙주 세포를 감염시킬 수 있는 능력을 제공한다. 지질 복합체(lipid complexes), 리포솜(liposomes) 및 덴드리머(dendrimers)와 같은 그 밖의 방법을 또한 사용할 수 있다. 본 분야에서의 기술분야의 이러한 것들은 이러한 또는 그 밖의 방법을 사용하여 목적하는 서열을 갖는 숙주 세포를 감염시킬 수 있다.
벡터는 염색체 복제[예를 들어, 플라스미드, 염색체 외 요소(extrachromosomal element), 미니염색체(minichromosome), 또는 인공적인 염색체]에 독립적인 복제, 염색체 외 독립체(extrachromosomal entity)로서 존재하는 독자적으로 복제하는 벡터(autonomously replicating vector)일 수 있다. 벡터는 자기-복제(self-replication) 확인하기 위한 어떠한 수단을 포함할 수 있다. 그 대신에, 숙주 내로 도입되었을 때, 벡터는 게놈 내에 통합되고, 이것이 통합된 것 내로 염색체와 함께 복제되었다. 더욱이, 숙주의 게놈 내로 도입될 수 있는 전체 DNA 또는 전이인자(transposon)를 함께 포함하는, 단일 벡터 또는 플라스미드 또는 둘 또는 그 이상의 벡터 또는 플라스미드를 사용할 수 있다.
벡터는 형질전환된 숙주(transformed hosts)의 쉬운 선택(easy selection)을 허용하는 하나 또는 그 이상의 선택가능한 마커(selectable markers)를 바람직하게 포함한다. 선택가능한 마커는, 예를 들어 살생제(biocide) 또는 바이러스성 저항(viral resistance), 중금속(heavy metals)에 대한 저항, 영양요구체(auxotrophs)에 대한 프로토트로피(prototrophy) 및 그 밖의 것과 같은 것의 제공에 의한 유전자 생성물이다. 박테리아 세포의 선택은 amp, gpt, neo 및 hyg 유전자와 같은 유전자에 의해 부여되는 길항미생물 저항성(antimicrobial resistance)을 기반으로 할 수 있다.
효모 숙주에 대한 적절한 마커(markers)는 예를 들어, ADE2, HIS3, LEU2, LYS2, MET3, TRP1 및 URA3 이다. 곰팡이 숙주(filamentous fungal host)에 사용되기 위한 선택적인 마커는, amdS[아세타미다아제(acetamidase)], argB[오르니틴 카르바모일트랜스페라아제(ornithine carbamoyltransferase)], bar[포스리노리친 아세틸트랜스퍼라아제(phosphinothricin acetyltransferase)], hph[히그로마이신 포스포트랜스페라아제(hygromycin phosphotransferase)], niaD[니트레이트 환원효소(nitrate reductase)], pyrG[오로티딘-5'-포스페이트 디카르복실라아제(orotidine-5'-phosphate decarboxylase)], sC[술페이트 아데닐트랜스페라아제(sulfate adenyltransferase)], 및 trpC[안트라닐산 생성효소(anthranilate synthase)]를 포함하지만, 이로 제한되지 않는다.
벡터는, 게놈의 독립된 세포에서 벡터의 자율증식(autonomous replication) 또는 숙주의 게놈 내로 벡터의 통합(integration)을 허용하는 요소를 바람직하게 포함한다.
숙주 게놈 내로의 통합을 위해, 벡터는 상동의(homologous) 또는 비상동의 재조합(nonhomologous recombination)에 의해 게놈 내로 벡터의 통합을 위한 벡터의 모든 그 밖의 요소(element) 또는 유전자의 서열을 필요로 할 수 있다. 그 대신에, 벡터는 숙주의 게놈 내로 상동의 재조합에 의해 통합을 지시하기 위해 추가적인 뉴클레오티드 서열을 포함할 수 있다. 추가적인 뉴클레오티드 서열은 벡터가 염색체에서 정확한 위치(precise location)에서의 숙주 게놈 내로 통합될 수 있게 한다. 정확한 위치에서의 통합의 가능성(likelihood)을 증가시키기 위해, 통합의 요소(integrational elements)는 바람직하게 충분한 수의 핵산, 100 내지 10,000 개의 염기쌍, 바람직하게 400 내지 10,000 개의 염기쌍 및 가장 바람직하게 800 내지 10,000 개의 염기쌍을 바람직하게 포함하고, 이는 상동의 재조합(homologous recombination)의 개연성(probability)을 향상시키기 위해서, 이에 상응하는 목표 서열(corresponding target sequence)을 갖는 높은 상동성을 갖는다. 통합의 요소(integrational elements)는 숙주의 게놈에서 목표 서열을 갖는 상동적인 모든 서열일 수 있다. 게다가, 통합의 요소는 비-코딩(non-encoding) 또는 코딩 뉴클레오티드 서열일 수 있다. 반면에, 벡터는 비-상동성(non-homologous) 재조합에 의해 숙주의 게놈 내로 통합될 수 있다.
자율증식(autonomous replication)에 대해서, 벡터는 논의되고 있는 숙주에서 벡터가 자체적으로 복제할 수 있는 복제 원점(origin of replication)을 추가로 포함할 수 있다. 복제 원점은 세포에서의 작용하는 자율증식을 중재하는(mediating) 모든 플라스미드 복제인자(plasmid replicator) 일 수 있다. 용어 "복제 원점" 또는 "플라스미드 복제인자" 는 플라스미드 또는 벡터가 생체 내에서 복제할 수 있는 서열로서 나타낸 것이다. 효모 숙주에서 사용을 위한 복제 원점의 예는 복제의 2 미크론 오리진(micron origin), ARS1, ARS4, ARS1 및 CEN3 의 조합 및 ARS4 및 CEN6 의 조합이다. 곰팡이 세포에서 유용한 복제 원점의 예는 AMA1 및 ANSI 이다(Gems et al ., 1991; Cullen et al ., 1987; WO 00/24883). AMA1 유전자 및 유전자를 포함하는 플라스미드 또는 벡터의 구조(construction)의 분리는 WO 00/24883 에 나타낸 방법에 따라 성취할 수 있다.
그 밖의 숙주에 대해서, 형질전환의 절차는, 예를 들어 클루이베르마이세스에 대해서 in Jeremiah D. Read, et al ., Applied and Environmental Microbiology, Aug. 2007, p. 5088-5096, 자모모나스에 대해서 in Osvaldo Delgado, et al ., FEMS Microbiology Letters 132, 1995, 23-26, 피치아 스티피트 스에 대해서 US 7,501,275 및 클로스트리디움에 대해서 WO 2008/040387 에 기재되어 있다.
유전자의 하나 보다 많은 복제는, 유전자 산물의 생산을 증가시키기 위해 숙주 내로 삽입할 수 있다. 유전자의 복제 수(copy number)의 증가는 숙주 게놈 내로 유전자의 적어도 하나의 추가적인 복제를 통합시키거나, 또는 세포가 선택가능한 마커의 증폭된 복제를 포함하는 경우 뉴클레오티드 서열을 갖는 증폭가능한 선택가능한 마커 유전자(amplifiable selectable marker gene)를 포함시켜서 수득할 수 있고, 이에 의해서 유전자의 추가적인 복제(additional copies)는 적절한 선택가능한 유전자의 존재 하에서 세포를 배양시켜 선택할 수 있다.
본 발명의 재조합 발현 벡터를 구성하기 위해 상기한 요소를 묶는데(ligate) 사용하는 절차는 본 분야에서의 숙련자에서 널리 공지되어 있다(예를 들어, Sambrook et al., 1989, supra 참조).
숙주 세포는 적어도 하나의 발현 벡터와 함께 변형된다. 오직 하나의 발현벡터를 사용하였을 때[중간물질(intermediate)의 첨가 없이], 벡터는 필요한 핵산 서열의 모두를 포함할 수 있다.
숙주 세포를 발현 벡터로 변형시키자마자, 숙주 세포는 성장이 가능하다. 본 발명의 방법은 세포에서 재조합 핵산이 발현된 것과 같이 숙주 세포를 배양하는 것을 포함할 수 있다. 미생물의 숙주에 대해서, 이러한 과정은 적절한 배지에서 세포의 배양을 수반한다. 일반적인 세포는 적절한 배지에서 35 ℃ 에서 성장한다. 본 발명의 바람직한 성장 배지는, 예를 들어 Luria Bertani(LB) 브로스(broth), Sabouraud Dextrose (SD) 브로스 또는 효모 배지(Yeast medium, YM) 브로스와 같은 상업적으로 바람직한 배지를 포함한다. 그 밖의 특정되거나 합성된 성장 배지를 또한 사용할 수 있고, 특정 숙주 세포의 성장을 위한 적절한 배지는 미생물 또는 발효 과학의 분야에서의 숙련자에게 공지될 것이다. 성장을 위한 적절한 온도 범위 및 그 밖의 조건은 본 분야에 공지되어 있다(예를 들어, Bailey and Ollis 1986 참조).
본 발명의 몇몇의 양상에 따라, 배양 배지는 숙주 세포에 대한 탄소원(carbon source)을 포함한다. 이러한 "탄소원"은 원핵 또는 단순한 진핵 세포의 성장을 위한 탄소원으로서 사용된 적절한 화합물 또는 기질을 일반적으로 나타낸다. 탄소원은, 중합체, 탄수화물, 산, 알코올, 알데히드, 케톤, 아미노산, 펩티드 등을 포함하지 이로 제한되지 않는 다양한 형태일 수 있다. 이러한 것은, 글루코스, 올리고당, 다당류와 같은 다양한 단량체, 셀룰로스 또는 헤미셀룰로스와 같은 바이오매스, 자일로스, 아라비노스, 수크로스와 같은 이당류, 포화 또는 불포화 지방산, 숙시네이트(succinate), 락테이트(lactate), 아세테이트(acetate), 에탄올 등을 포함한다. 탄소원은 글루코스를 포함하지만 이로 한정되지 않는 추가적인 광합성(photosynthesis) 산물일 수 있다.
바람직한 실시형태에서, 탄소원은 셀룰로스 또는 헤미셀룰로스와 같은 바이오매스 중합체이다. 본원에 기재된 바와 같은 "바이오매스 중합체" 는 생물학적인 물질(biological material)을 포함하는 모든 중합체이다. 생물학적인 물질은 살아있거나 죽은 것일 수 있다. 바이오매스 중합체는, 예를 들어 셀룰로스, 자일란, 자일로스, 헤미셀룰로스, 리그닌, 만난 및 바이오매스에서 일반적으로 발견되는 그 밖의 물질을 포함한다. 바이오매스 중합체의 근원(sources)의 비-제한적인 예는, 풀(grasses)[예를 들어, 지팽이풀(switchgrass), 미스칸터스( Miscanthus )], 왕겨(rice hull), 버개스(bagasse), cotton, jute, hemp, 아마(flax), 대나무(bamboo), 사이잘(sisal), 마닐라삼섬유(abaca), 짚(straw), 잎(leaves), 잔디 부스러기(grass clippings), 옥수수대(corn stover), 옥수수 속대(corn cobs), 증류립(distillers grains), 콩과 식물(legume plants), 수수(sorghum), 사탕수수(sugar cane), 사탕무 펄프(sugar beet pulp), 우드칩(wood chips), 톱밥(sawdust) 및 바이오매스 크롭(biomass crops)[예를 들어, 크람베( Crambe )]를 포함한다.
적절한 탄소원에 더하여, 배지는 본 분야에서 숙련자에게 공지되고, 배양물의 성장 및 다양한 당의 발효에 필수적인 효소적인 경로(enzymatic pathways)의 프로모션(promotion), 및 탄화수소 및 탄화수소 유도체의 생산에 적절한, 무기물, 염, 보조인자, 완충용액 및 그 밖의 성분을 포함해야한다. 반응물은, 호기성(aerobic), 산소 결핍(anoxic) 또는 혐기성의 조건(anaerobic conditions)은 미생물의 요구를 기반으로한 바람직한, 호기성 또는 혐기성 조건 하에서 반응이 실시될 수 있다. 숙주 세포가 성장하고/성장하거나 증가됨에 따라, 다양한 당 또는 바이오매스 중합체에서의 성장, 당 발효, 또는 탄화수소 또는 탄화수소 유도체의 합성에 필요한 효소, 운반체 또는 그 밖의 단백질의 발현은 영향을 받는다.
세포 내로 당의 수송을 증가시키는 방법
본 발명은 세포 내로 당의 수송을 증가시키는 방법을 제공한다. 하나의 양상에서, 본 발명은, 숙주 세포를 준비하는 첫 번째 단계를 포함하는 세포 내로 셀로덱스트린을 수송하는 방법을 제공하고, 상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 하기의 하나 또는 그 이상은 사실이다 : 트랜스멤브레인 α-헬릭스 1 은 SEQ ID NO: 1 을 포함하고, 트랜스멤브레인 α-헬릭스 2 는 SEQ ID NO : 2 를 포함하고, 트랜스멤브레인 α-헬릭스 2 및 트랜스멤브레인 α-헬릭스 3 을 연결하는 루프는 SEQ ID NO : 3 을 포함하고, 트랜스멤브레인 α-헬릭스 5 는 SEQ ID NO : 4 를 포함하고, 트랜스멤브레인 α-헬릭스 6 은 SEQ ID NO : 5 를 포함하고, 트랜스멤브레인 α-헬릭스 6 및 트랜스멤브레인 α-헬릭스 7 사이의 서열은 SEQ ID NO : 6 을 포함하고, 트랜스멤브레인 α-헬릭스 7 은 SEQ ID NO : 7 을 포함하고, 트랜스멤브레인 α-헬릭스 10 및 트랜스멤브레인 α-헬릭스 11 및 이들 사이의 서열은 SEQ ID NO : 8 을 포함한다. 본 방법은 재조합 폴리뉴틀레오티드가 발현되는 것과 같은 세포를 배양하는 두 번째 단계를 포함하고, 재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로 셀로덱스트린의 수송이 증가되었다. 세포 내로의 셀로덱스트린의 수송은 셀로비오스가 단일 탄소원(sole carbon source)인 경우, S. 세레비시아 숙주 세포의 능력을 측정하거나 또는 세포 내로 [3H]-셀로비오스의 흡수를 측정하는 것과 같은 실시예 9 에서 기재된 이러한 방법을 포함하는, 본 분야에서 숙련자에게 알려진 모든 방법을 측정할 수 있다. 일반적으로, 재조합 폴리뉴클레오티드를 포함하는 숙주 세포 및 재조합 폴리뉴클레오티드를 포함하지 않는 숙주 세포는, 그외의 유전적인 백그라운드(genetic background)에서 동일하다.
특정 실시형태에서, 폴리펩티드는 NCU00801 또는 NCU08114 와 적어도 29 %, 적어도 30 %, 적어도 35 %, 적어도 40 %, 적어도 45 %, 적어도 50 %, 적어도 55 %, 적어도 60 %, 적어도 65 %, 적어도 70 %, 적어도 75 %, 적어도 80 %, 적어도 85 %, 적어도 90 %, 적어도 95 %, 적어도 100 % 의 의 아미노산 동일성을 갖는다. 특정 실시형태에서, 숙주 세포는 β-글루코시다아제의 촉매 도메인을 코딩하는 재조합 폴리뉴클레오티드를 또한 포함한다. 이러한 실시형태는 셀로덱스트린을 사용하기 위한 내인성 능력(endogenous ability)이 결핍된 숙주 세포에 유용하다. 바람직하게, β-글루코시다아제의 촉매 도메인은 세포 내이다. 바람직한 실시형태에서, β-글루코시다아제는 네우로스포라 크라사로부터 온 것이다. 특히 바람직한 실시형태에서, β-글루코시다아제는 NCU00130 에 의해 코드된다.
세포 내로 셀로덱스트린의 수송을 증가시키는 방법에 있어서, 상기 세포는 변경된 유기체(altered organism)로부터 셀룰라아제-함유 효소 혼합물(cellulase-containing enzyme mixture)을 포함하는 배지에서 배양시킬 수 있고, 상기 혼합물은 변경된 유기체로부터 셀룰라아제-함유 혼합물과 비교하여 감소된 β-글루코시다아제 활성도를 갖는다. 유기체는, β-글루코시다아제를 코딩하는 유전자의 돌연변이 또는 목표된 RNA 간섭(targeted RNA interference) 또는 이와 같은 것과 같은 β-글루코시다아제의 발현을 감소시키기 위해 변경시킬 수 있다.
또 다른 양상에서, 본 발명은, 하기의 단계를 포함하는 세포 내로 자일로스의 수송을 증가시키는 방법을 제공하고, NCU00821 또는 STL12/XUT6 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 준비하는 단계, 및 재조합 폴리뉴클레오티드가 발현된 것과 같은 세포를 배양하는 단계를 포함하되, 상기 재조합 폴리뉴클레오티드의 발현으로, 재조합 폴리뉴클레오티드가 포함되지 않는 세포와 비교하여 세포 내로 자일로스의 수송을 증가시킨다. 또 다른 양상에서, 본 발명은 하기의 단계를 포함하는, 세포 내로 아라비노스의 수송을 증가시키는 방법을 제공하고, XUT1 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하는 단계, 및 재조합 폴리뉴클레오티드가 발현되는 세포를 배양하는 단계를 포함하되, 상기 재조합 폴리뉴클레오티드의 발현으로, 재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여 세포 내로 아라비노스의 수송을 증가시킨다. 또 다른 양상에서, 본 발명은 하기의 단계를 포함하는, 세포 내로 아라비노스 또는 글루코스의 수송을 증가시키는 방법을 제공하고, NCU06138 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 준비하는 단계, 및 재조합 폴리뉴클레오티드가 발현되는 세포를 배양하는 단계를 포함하되, 재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로 아라비노스 또는 글루코스의 수송을 증가시킨다. 또 다른 양상에서, 본 발명은 다음 단계를 포함하는, 세포 내로 자일로스 또는 글루코스의 수송을 증가시키는 방법을 제공하고, UT2, SUT3 또는 XUT3 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 준비하는 단계, 및 재조합 폴리뉴클레오티드가 발현되는 세포를 배양하는 단계를 포함하되, 재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로 자일로스 또는 글루코스의 수송을 증가시킨다. 또 다른 양상에서, 본 발명은 다음 단계를 포함하는, 세포 내로 자일로스, 아라비노스 또는 글루코스의 수송을 증가시키는 방법을 제공하고, NCU04963 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 준비하는 단계, 및 재조합 폴리뉴클레오티드가 발현되는 세포를 배양하는 단계를 포함하되, 재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로 자일로스, 아라비노스 또는 글루코스의 수송을 증가시킨다.
세포 내로의 자일로스, 아라비노스 또는 글루코스의 수송은 실시예 11 에 기재된 방법을 포함하는 본 분야의 숙련자에게 공지된 모든 방법으로 측정될 수 있다. 이러한 방법은, 예를 들어 삼투압(osmosis) 및 고성능 액체 크로마토그래피(high performance liquid chromatography)를 사용한 분석으로 숙주 세포로부터 축척된 D-자일로스 및 자일리톨 또는 L-아라비노스 및 아라비니톨의 추출에 의한 D-자일로스 또는 L-아라비노스를 측정하고, 단일 탄소원으로서 글루코스에서 성장할 수 있는 능력이 결핍된(lacking) 숙주 세포를 사용한 글루코스 수송을 측정하는 것을 포함한다. 일반적으로, 재조합 폴리뉴클레오티드를 포함하는 숙주 세포 및 재조합 폴리뉴클레오티드를 포함하지 않는 숙주 세포는, 그외의 유전적인 백그라운드(genetic background)에서 동일하다.
특정 실시형태에서, 숙주 세포는, 하나 또는 그 이상의 재조합 폴리뉴클레오티드를 또한 포함하되, 상기 하나 또는 그 이상의 재조합 폴리뉴클레오티드는 오탄당 이용에 포함되는 하나 또는 그 이상의 효소를 코드한다. 하나 또는 그 이상은, 예를 들어 L-아라비노스 이성질화효소, L-리블로인산화효소, L-리블로스-5-P 4 에피머화효소, 자일로스 이성질화효소, 자일루로키나아제, 알도스 환원효소, L-아라비니톨 4-탈수소효소, L-자일루로스 환원효소 및 자일리톨 탈수소효소 또는 본 분야에서 공지된 그 밖의 오탄당 이용 효소일 수 있다.
세포의 성장을 증가시키는 방법
본 발명은 세포의 성장을 증가시키는 방법을 제공한다. 하나의 양상에서, 본 발명은, 숙주 세포를 준비하는 첫 번째 단계를 포함하는, 세포의 성장을 증가시키는 방법을 제공하고, 상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 하기의 하나 또는 그 이상은 사실이다 : 트랜스멤브레인 α-헬릭스 1 은 SEQ ID NO: 1 을 포함하고, 트랜스멤브레인 α-헬릭스 2 는 SEQ ID NO : 2 를 포함하고, 트랜스멤브레인 α-헬릭스 2 및 트랜스멤브레인 α-헬릭스 3 을 연결하는 루프는 SEQ ID NO : 3 을 포함하고, 트랜스멤브레인 α-헬릭스 5 는 SEQ ID NO : 4 를 포함하고, 트랜스멤브레인 α-헬릭스 6 은 SEQ ID NO : 5 를 포함하고, 트랜스멤브레인 α-헬릭스 6 및 트랜스멤브레인 α-헬릭스 7 사이의 서열은 SEQ ID NO : 6 을 포함하고, 트랜스멤브레인 α-헬릭스 7 은 SEQ ID NO : 7 을 포함하고, 트랜스멤브레인 α-헬릭스 10 및 트랜스멤브레인 α-헬릭스 11 및 이들 사이의 서열은 SEQ ID NO : 8 을 포함하고, 폴리펩티드는 셀로덱스트린을 수송한다. 본 방법은 셀로덱스트린을 포함하는 배지에서의 숙주 세포를 배양하는 두 번째 단계를 포함하고, 상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 보다 빠른 비율로 성장한다. 숙주 세포의 성장 비율은 본 분야에서의 숙련자에게 공지된 방법에 의해 측정될 수 있다. 일반적으로, 세포의 성장 비율은 광학 밀도(optical density)에 의한 현탁액(suspension)에서의 세포의 농도를 평가하여서 측정할 수 있다. 바람직하게, 재조합 폴리뉴클레오티드를 포함하는 숙주 세포 및 재조합 폴리뉴클레오티드를 포함하지 않는 숙주 세포는, 그외의 유전적인 백그라운드(genetic background)에서 동일하다. 셀로덱스트린을 포함하는 배지는 셀룰로스와 같은 바이오매스 중합체의 효소적인 처리로 인한다.
특정 실시형태에서, 폴리펩티드는 NCU00801 또는 NCU08114 와 적어도 29 %, 적어도 30 %, 적어도 35 %, 적어도 40 %, 적어도 45 %, 적어도 50 %, 적어도 55 %, 적어도 60 %, 적어도 65 %, 적어도 70 %, 적어도 75 %, 적어도 80 %, 적어도 85 %, 적어도 90 %, 적어도 95 %, 적어도 99 %, 적어도 100 % 의 아미노산 동일성을 갖는다. 특정 실시형태에서, 숙주 세포는 β-글루코시다아제의 촉매 도메인을 코딩하는 재조합 폴리뉴클레오티드를 또한 포함한다. 이러한 실시형태는 셀로덱스트린을 이용하기 위해 내인성 능력(endogenous ability)이 결핍된(lacking) 숙주 세포에 유용한다. 바람직하게, β-글루코시다아제의 촉매 도메인은 세포내이다. 바람직한 실시형태에서, β-글루코시다아제는 네우로스포라 크라사로부터 온 것이다. 특히 바람직한 실시형태에서, β-글루코시다아제는 NCU00130 에 의해 코드된다.
세포의 성장을 증가시키는 방법에 있어서, 배양하는 배지는 변경된 유기체로부터 셀룰라아제-함유 효소 혼합물을 포함하고, 상기 혼합물을 변경되지 않는 유기체로부터의 셀룰라아제-함유 혼합물과 비교하여 감소된 β-글루코시다아제 활성도를 갖는다. 유기체는 목표하는 RNA 간섭(targeted RNA interference) 또는 이와 같은 것에 의하거나 또는 β-글루코시다아제를 코딩하는 유전자의 돌연변이와 같은, β-글루코시다아제의 발현을 감소시키기 위해 변경시켰다.
또 다른 양상에서, 본 발명은 하기의 단계를 포함하는, 세포의 성장을 증가시키는 방법을 제공하고, 자일로스를 수송하는 NCU00821 또는 STL12/XUT6 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하는 단계, 및 자일로스를 포함하는 배지에서 숙주 세포를 배양하는 단계를 포함하되, 상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 더 빠른 비율로 성장한다. 또 다른 양상에서, 본 발명은 하기의 단계를 포함하는, 세포의 성장을 증가시키는 방법을 제공하고, 아라비노스를 수송하는 XUT1 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하는 단계, 및 아라비노스를 포함하는 배지에서 숙주 세포를 배양하는 단계를 포함하되, 상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 더 빠른 비율로 성장한다. 또 다른 양상에서, 본 발명은 하기의 단계를 포함하는, 세포의 성장을 증가시키는 방법을 제공하고, 아라비노스 및 글루코스를 수송하는 NCU06138 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하는 단계, 및 아라비노스 또는 글루코스를 포함하는 배지에서 숙주 세포를 배양하는 단계를 포함하되, 상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 더 빠른 비율로 성장한다. 또 다른 양상에서, 본 발명은 하기의 단계를 포함하는, 세포의 성장을 증가시키는 방법을 제공하고, 자일로스 및 글루코스를 수송하는 SUT2, SUT3 또는 XUT3 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하는 단계, 및 자일로스 또는 글루코스를 포함하는 배지에서 숙주 세포를 배양하는 단계를 포함하되, 상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 더 빠른 비율로 성장한다. 또 다른 양상에서, 본 발명은 하기의 단계를 포함하는, 세포의 성장을 증가시키는 방법을 제공하고, 자일로스, 아리비노스 및 글루코스를 수송하는 NCU04963 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하는 단계, 및 자일로스, 아라비노스 또는 글루코스를 포함하는 배지에서 숙주 세포를 배양하는 단계를 포함하되, 상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 더 빠른 비율로 성장한다.
숙주 세포의 성장 비율은 본 분야의 숙련자에서 공지된 모든 방법으로 측정될 수 있다. 일반적으로, 세포의 성장 비율은 공학 밀도에 의한 현탁액에서의 세포 농도를 평가하여 측정할 수 있다. 바람직하게, 재조합 폴리뉴클레오티드를 포함하는 숙주 세포 및 재조합 폴리뉴클레오티드를 포함하지 않는 숙주 세포는, 그외의 유전적인 백그라운드(genetic background)에서 동일하다. 자일로스 또는 아라비노스를 포함하는 배지는 헤미셀룰로스와 같은 바이오매스 중합체의 산 처리에 로부터 기인한다. 글루코스를 포함하는 배지는 셀룰로스와 같은 바이오매스 중합체의 효소적 처리(enzymatic treatment)로부터 기인한다.
특정 실시형태에서, 숙주 세포는 하나 또는 그 이상의 재조합 폴리뉴클레오티드를 또한 포함하되, 상기 하나 또는 그 이상의 폴리뉴클레오티드는 오탄당 이용에 포함되는 하나 또는 그 이상의 효소를 코드한다. 상기 하나 또는 그 이상의 효소는, 예를 들어 L-아라비노스 이성질화효소, L-리블로인산화효소, L-리블로스-5-P 4 에피머화효소, 자일로스 이성질화효소, 자일루로키나아제, 알도스 환원효소, L-아라비니톨 4-탈수소효소, L-자일루로스 환원효소 및 자일리톨 탈수소효소 또는 본 분야에서 공지된 그 밖의 오탄당 이용 효소일 수 있다.
하나의 양상에서, 본 발명은 바이오매스 중합체에서 세포의 성장을 증가시키는 방법을 제공한다. 바람직한 실시형태에서, 바이오매스 중합체는 셀룰로스이다. 또 다른 바람직한 실시형태에서, 바이오매스 중합체는 헤미셀룰로스이다. 본 발명의 하나의 양상에 따라, 본 발명은 NCU07705 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 준비하는 것을 포함한다. 본 발명의 또 다른 양상에 따라, 본 방법은 바이오매스 중합체를 포함하는 배지에서 세포를 배양하는 것을 포함하고, 상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포 보다 배지에서 더 빠른 비율로 성장하였다.
본 발명의 또 다른 양상에서, 본 발명은, 하기의 단계를 포함하는, 세포의 성장을 증가시키는 방법을 제공하고, NCU01517, NCU09133 또는 NCU10040 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하는 단계, 및 헤미셀룰로스를 포함하는 배지에서 세포를 배양하는 단계를 포함하되, 상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 더 빠른 비율로 성장하였다.
본 발명의 또 다른 양상에 따라, 본 방법은 NCU05137 폴리펩티드를 코드하는 내인성 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하는 것을 포함한다. 본 발명의 또 다른 양상에 따라, 본 발명은 내인성 폴리뉴클레오티드의 발현을 저해하고, 바이오매스 중합체를 포함하는 배지에서 세포를 배양하는 것을 포함하되, 상기 숙주 세포는, 내인성 폴리뉴클레오티드의 발현이 저해되지 않는, 세포보다 배지에서 더 빠른 비율로 성장하였다.
본 발명의 방법은 세포에서 재조합 핵산이 발현되는 숙주 세포를 배양하는 것을 포함한다. 미생물 숙주로부터, 상기 과정은 적절한 배지에서 세포를 배양하는 것을 수분한다. 일반적인 세포는 적절한 배지에서 35 ℃ 에서 성장시켰다. 본 발명에서 바람직한 성장 배지는, 예를 들어 Luria Bertani (LB) 브로스(broth), Sabouraud Dextrose (SD) 브로스 또는 효소 배지(YM) 브로스와 같은 일반적으로 상업적으로 제조된 배지를 포함한다. 그 밖의 특정되거나 합성된 성장 배지를 또한 사용할 수 있고, 특정한 숙주 세포의 성장에 적절한 배지는, 미생물학 또는 발효과학의 분야에서 숙련자에게 널리 공지되어 있다. 성장에 적절한 온도 범위 및 그 밖의 조건은 본 분야에서 공지되어 있다(예를 들어, Bailey and Ollis 1986 참조)
배지에서의 중합체의 근원은, 예를 들어 풀(grasses)[예를 들어, 지팽이풀(switchgrass), 미스칸터스( Miscanthus )], 왕겨(rice hull), 버개스(bagasse), 무명(cotton), 주트(jute), 대마(hemp), 아마(flax), 대나무(bamboo), 사이잘(sisal), 마닐라삼섬유(abaca), 짚(straw), 잎(leaves), 잔디 부스러기(grass clippings), 옥수수대(corn stover), 옥수수 속대(corn cobs), 증류립(distillers grains), 콩과 식물(legume plants), 수수(sorghum), 사탕수수(sugar cane), 사탕무 펄프(sugar beet pulp), 우드칩(wood chips), 톱밥(sawdust) 및 바이오매스 크롭(biomass crops)[예를 들어, 크람베( Crambe )]를 포함한다. 바이오매스 중합체에 추가적으로, 배지는, 배양물의 성장에 적절한, 무기질, 염, 공동인자(cofactors), 완충용액 및 본 분야의 숙련자에게 널리 알려진 그 밖의 성분을 포함해야한다. 숙주 세포의 성장 비율은 본 분야에서의 숙련자에서 공지된 모든 방법으로 측정할 수 있다.
본 발명의 특정 실시형태에서, 셀룰라아제의 발현은 재조합 폴리뉴클레오티드의 발현 하상에서 숙주 세포에서 증가되었다. 본원에서 사용된 "셀룰라아제"는, 셀룰로스 중합체를 보다 더 짧은 셀로-올리고당 올리고머(shorter cello-oligosaccharide oligomers), 셀로비오스 및/또는 당으로 가수분해할 수 있는 효소의 카테고리(category)를 나타낸 것이다. 셀룰라아제는 엑소글루카나아제(exoglucanases), 엑소셀로비오히드롤라아제(exocellobiohydrolases), 엔도글루카나아제(endoglucanases) 및 글루코시다이제를 포함하지만 이로 한정되지 않는다. 셀룰라아제의 발현은 본 분야에서 공지된 RP-PCR 또는 그 밖의 방법으로 측정될 수 있다.
본 발명의 특정 실시형태에서, 헤미셀룰라아제의 발현은 재조합 폴리뉴클레오티드의 발현 하에서 숙주 세포에서 증가되었다. 본원에 기재된 "헤미셀룰라아제"는 헤미셀룰로스 중합체를 가수분해하는 효소의 카테고리를 나타낸 것이다. 헤미셀룰라아제는, 자일라나아제(xylanases), 만나나아제(mannanases), 아라비나아제(arabinases)[둘 다 엔도 및 엑소 종류(endo and exo kinds)] 및 이들과 상응하는 글리코시다아제를 포함하지만 이로 제한되지 않는다. 헤미셀룰라아제의 발현은 RT-PCR 또는 본 분야에서 공지된 그 밖의 방법으로 측정할 수 있다.
내인성 폴리뉴클레오티드의 발현의 저해는, 예를 들어 유전자의 기능 또는 유전자의 산물(예를 들어, 유전자에 의해 코드되는 단백질)의 기능에서의 감소로 인한 유전적 변형에 의해 성취될 수 있고, 이는 유전자의 발현의 불활성화(완전한 또는 부분적인), 결실, 중단(interruption), 장애(blockage), 침묵(silencing), 하향-조절(down-regulation) 또는 감쇠(attenuation)로서 나타낸 것일 수 있다. 예를 들어, 이러한 유전자에 의해 코드되는 단백질의 기능에서의 감소를 야기하는 유전자에서의 유전적인 변형은, 유전자의 완전한 결실(즉, 유전자가 존재하지 않고, 따라서 단백질이 존재하지 않는다), 단백질의 불완전하거나 또는 번역이 없는 것을 야기하는 유전자에서의 돌연변이(예를 들어, 단백질이 발현되지 않는다), 또는 단백질의 천연적인 기능을 감소시키거나 폐지하는(abolish) 유전자에서의 돌연변이(예를 들어, 효소적인 활성도 또는 작용이 없거나 감소된 단백질이 발현된다)의 결과일 수 있다. 보다 명확하게, 본원에 기재된 단백질의 작용을 감소시키는 언급은, 숙주 세포에서 모든 유전적인 변형에 대해서 일반적으로 나타내었고, 단백질의 감소된 발현 및/또는 기능성(생물학적 활성도)을 야기하고, 단백질의 감소된 활성도(예를 들어, 감소된 수송), 단백질의 증가된 저해 또는 분해 뿐만 아니라 단백질의 발현의 감소 또는 저해를 포함하는 숙주 세포에서의 어떠한 유전적인 변형을 나타낸 것이다. 예를 들어, 본 발명의 단백질의 작용 또는 활성도는 단백질 생산의 저지(blocking) 또는 감소, 단백질 작용의 감소, 또는 단백질 작용의 저해에 의해 감소될 수 있다. 이러한 변형의 몇몇을 또한 조합할 수 있다. 단백질 생산의 저지(Blocking) 또는 감소는, 성장 배지에서 유도된 화합물의 존재를 필요로 하는 프로모터의 조절 하에서 단백질을 코딩하는 유전자를 배치하는 것을 포함할 수 있다. 유도물질(inducer)이 배지로부터 대폭 감소되는 것과 같은 조건을 설정함으로써, 단백질을 코딩하는 유전자의 발현(및 이에 따른 단백질의 합성)은 정지될 수 있다(turned off). 단백질의 작용을 저지하거나 감소시키는 것은 본원에서 참조문헌으로 인용된 U.S. Pat. No. 4,743,546 에 기재된 것과 유사한 제거 기술 접근법(excision technology approach)을 사용하는 것을 또한 포함할 수 있다. 이러한 접근법을 사용하기 위해, 흥미로운 단백질을 코딩하는 유전자는, 게놈으로부터 유전자가 특이적이고(specific), 조절된 제거(controlled excision)를 가능하게 하는 특이적인 유전적인 서열(specific genetic sequences) 사이에 클론되었다(cloned). 제거(Excision)는, 예를 들어 U.S. Pat. No. 4,743,546 에 기재된, 배양물의 배양 온도에서의 시프트(shift), 또는 몇몇 다른 물리적이거나(physical) 또는 영양상의 신호(nutritional signal)에 의해 유도된다(prompted).
본 발명의 특정 실시형태에서, 숙주 세포의 셀룰라아제 활성도는 내인성 폴리뉴클레오티드의 발현의 저해 상에서 증가되었다. 셀룰라아제 활성도는 실시예 5 에 기재된 바와 같은 방법 및 본 분야에서 알려진 그 밖의 방법으로 측정될 수 있다.
본 발명의 특정 실시형태에서, 숙주 세포의 헤미셀룰라아제 활성도는 내인성 폴리뉴클레오티드의 발현의 저해 상에서 증가되었다. 헤미셀룰라아제 활성도는 실시예 17 에 기재된 바와 같은 방법 및 본 분야에서 알려진 그 밖의 방법으로 측정될 수 있다.
공동-발효의 방법
본 발명의 하나의 양상은 셀룰로스-유도(derived) 및 헤미셀룰로스-유도 당을 공동-발효시키는(co-fermenting) 방법을 제공한다. 본원에서 사용된 바와 같이, 공동-발효(co-fermentation)는 동일한 베쎌(vessel)에서 하나 이상의 당이 숙주 세포에 의해 동시에 이용되는 것을 나타낸 것이다. 본 발명은, 셀로덱스트린 운반체를 코딩하는 첫 번째 재조합 폴리뉴클레오티드 및 β-글루코시다아제의 촉매 도메인을 코딩하는 두 번째 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하는 단계, 및 셀룰로스-유도 당 및 헤미셀룰로스-유도 당을 포함하는 배지에서 숙주 세포를 배양하는 단계를 포함하되, 상기 재조합 폴리뉴클레오티드의 발현은 셀룰로스-유도 당 및 헤미셀룰로스-유도 당을 공동-발효시킬 수 있다.
첫 번째 재조합 폴리뉴클레오티드는 세포 내로 셀로덱스트린을 수송할 수 있는 어떠한 폴리펩티드를 코드할 수 있다. 셀로덱스트린 수송은, 실시예 9 에 기재된 방법을 포함하는, 분 분야의 숙련자에게 공지된 방법으로 측정될 수 있다. 바람직한 실시형태에서, 첫 번째 재조합 폴리뉴클레오티드는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하고, 하기의 하나 또는 그 이상은 사실이다 : 트랜스맴브레인 α-헬릭스 1 은 SEQ ID NO: 1 을 포함하고, 트랜스맴브레인 α-헬릭스 2 는 SEQ ID NO: 2 를 포함하고, 트랜스맴브레인 α-헬릭스 2 와 트랜스맴브레인 α-헬릭스 3 를 연결하는 루프는 SEQ ID NO: 3 을 포함하고, 트랜스맴브레인 α-헬릭스 5 는 SEQ ID NO: 4 를 포함하고, 트랜스맴브레인 α-헬릭스 6 은 SEQ ID NO: 5 를 포함하고, 트랜스맴브레인 α-헬릭스 6 과 트랜스맴브레인 α-헬릭스 7 사이의 서열은 SEQ ID NO: 6 을 포함하고, 트랜스맴브레인 α-헬릭스 7 은 SEQ ID NO: 7 를 포함하고, 및 트랜스맴브레인 α-헬릭스 10 및 트랜스맴브레인 α-헬릭스 11 및 이들 사이의 서열은 SEQ ID NO: 8 을 포함한다. 이러한 폴리펩티드의 예는 NCU00801, NCU00809, NCU08114, XP_001268541.1 및 LAC2 를 포함한다. 바람직한 실시형태에서, 첫 번째 재조합 폴리펩티드는 NCU00801 를 코드한다.
두 번째 재조합 폴리뉴클레티드는, 글루코스를 방출하면서 β-D-글루코시드에서 말단의 비-환원된 잔기(terminal non-reducing residues)를 가수분해를 촉매하는 능력을 갖는 어떠한 촉매 도메인(any catalytic domain)을 코드할 수 있다. 바람직하게, β-글루코시다아제 촉매 도메인은 숙주 세포에서 세포 내에 위치한다. 특정 실시형태에서, β-글루코시다아제의 근원은 N. 크라사 β-글루코시다아제이다. 바람직한 실시형태에서, β-글루코시다아제 도메인의 근원은 NCU00130 이다. 상이한 근원으로부터의 촉매 도메인이 상이한 셀로덱스트린 운반체를 갖는 것이 최상으로 작업할 수 있다.
특정 실시형태에서, 숙주 세포는 하나 또는 그 이상의 재조합 폴리뉴클레오티드를 또한 포함하고, 상기 하나 또는 그 이상의 폴리뉴클레오티드는 오탄당 이용에 포함되는 하나 또는 그 이상의 효소를 코드한다. 선택적으로, 오탄당 이용에 포함되는 하나 또는 그 이상의 효소를 코딩하는 하나 또는 그 이상의 폴리뉴클레오티드는 숙주 세포에 대해서 내인성(endogenous)일 수 있다. 하나 또는 그 이상의 효소는, 예를 들어 L-아라비노스 이성질화효소, L-리블로인산화효소, L-리블로스-5-P 4 에피머화효소, 자일로스 이성질화효소, 자일루로키나아제, 알도스 환원효소, L-아라비니톨 4-탈수소효소, L-자일루로스 환원효소 및 자일리톨 탈수소효소 및 본 분야의 숙련자에게 공지된 그 밖의 오탄당-이용 효소를 포함한다.
특정 실시형태에서, 숙주 세포는 세 번째 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리뉴클레오티드는 오탄당 운반체를 코드한다. 선택적으로, 숙주 세포는 오탄당 운반체를 코딩하는 내인성 폴리뉴클레오티드를 포함할 수 있다. 특정 실시형태에서, 오탄당 운반체는 세포 내로 자일로스 및/또는 아라비노스를 수송한다. 특정 실시형태에서, 세 번째 재조합 폴리뉴클레오티드는 NCU00821, NCU04963, NCU06138, STL12/XUT6, SUT2, SUT3, XUT1 또는 XUT3 과 같은 폴리펩티드를 코드한다. 숙주 세포에서 오탄당 운반체의 발현은, 만약 오탄당 당 중에 존재하는 글루코스가 성장 배지라면, 공동-발효의 효율성은 증진될 것이다.
본원에 기재된 바와 같은 공동-발효의 방법에서, 셀룰로스-유도 당은 바람직하게 셀로비오스, 셀로트리오스 및 셀로테트라오스를 포함하고, 헤미셀룰로스-유도 당은 바람직하게 자일로스 및 아라비노스를 포함한다. 일반적으로, 숙주 세포에 의한 공동-발효를 위한 셀룰로스-유도 당 및 헤미셀룰로스-유도 당을 제조하기 위해, 리그노셀룰로스계 바이오매스(lignocellulosic biomass)는, 이의 구조를 변경시키기 위해 첫 번째로 예비처리하였고(pretreated), 셀룰로스의 보다 나은 효소적 가수분해(better enzymatic hydrolysis)를 가능하게 한다. 예비 처리는, 예를 들어 암모니아 파이버/냉동 폭발(ammonia fiber/freeze explosion), 칼슘 또는 수산화나트륨을 기반으로 한 라임방법(lime method), 및 산 촉매와 함께 또는 산 촉매 없는 증기 폭발(steam explosion)을 포함하는, 물리적이고 화학적인 방법을 포함할 수 있다. 산 처리는 리그노셀룰로스계 바이오매스의 헤미셀룰로스 성분으로부터의 자일로스 및 아라비노스를 방출할 것이다. 그 후에, 바람직하게, 예비처리된 바이오매스의 셀룰로스 성분은 셀룰라아제의 혼합물에 의해 가수분해되었다. 상업적으로 입수가능한 셀룰라아제 혼합물의 예는 Celluclast 1.5L®(Novozymes), Spezyme CP®(Genencor)(Scott W. Pryor, 2010, Appl Biochem Biotechnol) 및 Cellulyve 50L(Lyven)를 포함한다.
셀룰라아제 혼합물은 엔도글루카나아제, 엑소글루카나아제 및 β-글루코시다아제를 일반적으로 포함한다. 본원에 기재된 바와 같은 공동-발효의 방법에 있어서, 셀룰라아제 혼합물에서 β-글루코시다아제 활성도의 양은 가능한 많이 최소화되어야 한다. 예를 들어, 배양된 배지는 변경된 유기체로부터의 셀룰라아제-함유 효소를 포함할 수 있고, 상기 혼합물은 변경되지 않는 유기체로부터 셀룰라아제-함유 혼합물과 비교하여, 감소된 β-글루코시다아제 활성도를 갖는다. 유기체는, β-글루코시다아제를 코딩하는 유전자의 돌연변이 또는 목표하는 RNA 간섭(targeted RNA interference) 또는 이와 유사한 것과 같은 것에 의해 β-글루코시다아제의 발현을 감소시키도록 변경할 수 있다.
놀랍게도 실시예 17 에 기재된 바와 같이, 본 발명의 방법에 의한 셀로비오스 및 자일로스의 공동-발효는 숙주 세포에 의한 당 소비 및 에탄올 생산에 상승효과(synergistic effect)를 나타내었다.
탄화수소 또는 탄화수소 유도체의 합성 방법
본 발명의 하나의 양상은 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법을 제공한다.
본원에서 사용된 바와 같은 "탄화수소"는 전부 수소 및 탄소로 이루어진 유기 화합물이다. 탄화수소는, 메탄, 에탄, 에텐, 에틴(ethyne), 프로판, 프로펜, 프로핀(propyne), 시클로프로판, 알렌(allene), 부탄, 이소부텐(isobutene), 부텐, 부틴, 시클로부탄, 메틸시클로프로판, 부타디엔, 펜탄, 이소펜탄, 네오펜탄, 펜텐, 펜틴(pentyne), 시클로펜탄, 메틸시클로부탄, 에틸시클로프로판, 펜타디엔, 이소프레네, 헥산, 헥센, 헥신(hexyne), 시클로헥산, 메틸시클로펜탄, 에틸시클로부탄, 프로필시클로프로판, 헥사디엔, 헵테인(heptane), 헵텐(heptene), 헵틴(heptyne), 시클로헵테인, 메틸시클로헥산, 헵타디엔, 옥탄, 옥텐(octene), 옥틴(octyne), 시클로옥탄(cyclooctane), 옥타디엔(octadiene), 노난(nonane), 노넨(nonene), 노니엔(nonyne), 시클로노난(cyclononane), 노난디엔(nonadiene), 데칸, 데켄(decene), 데킨(decyne), 시클로데칸 및 데카디엔(decadiene)을 포함하지만 이로 제한되지 않는다.
본원에 사용된 바와 같은, "탄화수소 유도체"는 수소가 아닌 적어도 하나의 그 밖의 성분 및 탄소의 유기 화합물이다. 탄화수소 유도체는, 알코올(예를 들어, 아라비니톨, 부탄올, 에탄올, 글리세롤, 메탄올, 1,3-프로판디올, 소르비톨 및 자일리톨) ; 유기산(예를 들어, 아세트산, 아디프산, 아스코르브산, 시트르산, 2,5-디케토-D-글루콘산, 포름산, 푸마르산, 글루카르산, 글루콘산, 글루쿠론산(glucuronic acid), 글루타르산(glutaric acid), 3-히드록시프로피오닉산(hydroxypropionic acid), 이타콘산, 젖산, 말산, 말론산, 옥살산, 프로피온산, 숙신산 및 크실론산) ; 에스테르 ; 케톤(예를 들어, 아세톤) ; 알데히드(예를 들어, 푸르푸랄) ; 아미노산(예를 들어, 아스파르트산, 글루탐산, 글리신, 리신, 세린 및 트레오닌) ; 및 기체(gases)(예를 들어, 이산화탄소 및 일산화탄소)를 포함하지만, 이로 제한되지 않는다.
바람직한 실시형태에서, 탄화수소 또는 탄화수소 유도체는 연료로서 사용될 수 있다. 특히 바람직한 실시형태에서, 탄화수소 또는 탄화수소 유도체는 에탄올 또는 부탄올이다.
본 발명의 하나의 양상에 따라, 숙주 세포를 준비하는 첫 번째 단계를 포함하는 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법을 포함하고, 숙주 세포는 트랜스맴브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11 및 α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 상기 하나 또는 그 이상의 하기는 사실이다 : 트랜스맴브레인 α-헬릭스 1 은 SEQ ID NO: 1 을 포함하고, 트랜스맴브레인 α-헬릭스 2 는 SEQ ID NO: 2 를 포함하고, 트랜스맴브레인 α-헬릭스 2 와 트랜스맴브레인 α-헬릭스 3 를 연결하는 루프는 SEQ ID NO: 3 을 포함하고, 트랜스맴브레인 α-헬릭스 5 는 SEQ ID NO: 4 를 포함하고, 트랜스맴브레인 α-헬릭스 6 은 SEQ ID NO: 5 를 포함하고, 트랜스맴브레인 α-헬릭스 6 과 트랜스맴브레인 α-헬릭스 7 사이의 서열은 SEQ ID NO: 6 을 포함하고, 트랜스맴브레인 α-헬릭스 7 은 SEQ ID NO: 7 를 포함하고, 및 트랜스맴브레인 α-헬릭스 10 및 트랜스맴브레인 α-헬릭스 11 및 이들 사이의 서열은 SEQ ID NO: 8 을 포함하고, 상기 폴리펩티드는 탄화수소 또는 탄화수소 유도체의 합성을 위해 숙주 세포 내로 셀로덱스트린을 수송한다. 본 발명은 숙주 세포에 의해 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 셀로덱스트린 또는 셀로덱스트린의 근원을 포함하는 배지에서 숙주 세포를 배양하는 두 번째 단계를 포함하고, 세포 내로 셀로덱스트린의 수송은 재조합 폴리뉴클레오티드의 발현 상에서 증가되었다. 특정 실시형태에서, 폴리펩티드는, NCU00801 또는 NCU08114 와 적어도 29 %, 적어도 30 %, 적어도 35 %, 적어도 40 %, 적어도 45 %, 적어도 50 %, 적어도 55 %, 적어도 60 %, 적어도 65 %, 적어도 70 %, 적어도 75 %, 적어도 80 %, 적어도 85 %, 적어도 90 %, 적어도 95 %, 적어도 99 %, 적어도 100 % 의 아미노산 동일성을 갖는다. 특정 실시형태에서, 숙주 세포는 β-글루코시다아제의 촉매 도메인을 코딩하는 재조합 폴리뉴클레오티드를 또한 포함한다. 이러한 실시형태는 셀로덱스트린을 사용하기 위한 내인성 능력(endogenous ability)이 결핍된 숙주 세포에 대해서 유용하다. 바람직하게, β-글루코시다아제의 촉매 도메인은 세포 내이다. 바람직한 실시형태에서, β-글루코시다아제는 네우로스포라 크라사로부터 온 것이다. 특히 바람직한 실시형태에서, β-글루코시다아제는 NCU00130 에 의해 코드된다. 세포 내로 셀로덱스트린의 수송은 실시예 9 에 기재된 방법을 포함하는, 본 분야에서의 숙련자에게 공지된 어떠한 방법에 의해 측정될 수 있다. 일반적으로 셀로덱스트린의 근원은 셀룰로스이다.
배양 배지는 변경된 유기체로부터 셀룰라아제-포함 효소 혼합물을 포함할 수 있고, 상기 혼합물은 변경되지 않는 유기체로부터 셀룰라아제-포함 혼합물과 비교하여 감소된 β-글루코시다아제 활성도를 갖는다. 유기체는 β-글루코시다아제 를 코딩하는 유전자의 돌연변이 또는 목표하는 RNA 간섭 또는 이와 유사한 것과 같은, β-글루코시다아제의 발현을 저해하기 위해 변경될 수 있다.
본 발명의 또 다른 양상에 따라, 하기의 단계를 포함하는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법을 포함하고, 탄화수소 또는 탄화수소 유도체의 합성을 위한 숙주 세포 내로 자일로스를 수송하는 NCU00821 또는 STL12/XUT6 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는, 숙주 세포를 제공하는 단계, 및 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 자일로스 또는 자일로스의 근원을 포함하는 배지에서 숙주 세포를 배양하는 단계를 포함하되, 상기 세포 내로 자일로스의 수송은 재조합 폴리뉴클레오티드의 발현 상에서 증가되었다.
본 발명의 또 다른 양상에 따라, 하기의 단계를 포함하는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법을 포함하고, 탄화수소 또는 탄화수소 유도체의 합성을 위한 숙주 세포 내로 아라비노스를 수송하는 XUT1 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는, 숙주 세포를 제공하는 단계, 및 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 아라비노스 또는 아라비노스의 근원을 포함하는 배지에서 숙주 세포를 배양하는 단계를 포함하되, 상기 세포 내로 아라비노스의 수송은 재조합 폴리뉴클레오티드의 발현 상에서 증가되었다.
본 발명의 또 다른 양상에 따라, 하기의 단계를 포함하는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법을 포함하고, 탄화수소 또는 탄화수소 유도체의 합성을 위한 숙주 세포 내로 아라비노스 또는 글루코스를 수송하는 NCU06138 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는, 숙주 세포를 제공하는 단계, 및 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 아라비노스 또는 글루코스 또는 아라비노스 또는 글루코스의 근원을 포함하는 배지에서 숙주 세포를 배양하는 단계를 포함하되, 상기 세포 내로 아라비노스 또는 글루코스의 수송은 재조합 폴리뉴클레오티드의 발현 상에서 증가되었다.
본 발명의 또 다른 양상에 따라, 하기의 단계를 포함하는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법을 포함하고, 탄화수소 또는 탄화수소 유도체의 합성을 위한 숙주 세포 내로 자일로스 또는 글루코스를 수송하는 SUT2, SUT3 또는 XUT3 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는, 숙주 세포를 제공하는 단계, 및 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 자일로스 또는 글루코스 또는 자일로스 또는 글루코스의 근원을 포함하는 배지에서 숙주 세포를 배양하는 단계를 포함하되, 상기 세포 내로 자일로스 또는 글루코스의 수송은 재조합 폴리뉴클레오티드의 발현 상에서 증가되었다.
본 발명의 또 다른 양상에 따라, 하기의 단계를 포함하는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법을 포함하고, 탄화수소 또는 탄화수소 유도체의 합성을 위한 숙주 세포 내로 자일로스, 아라비노스 또는 글루코스를 수송하는 NCU04963 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는, 숙주 세포를 제공하는 단계, 및 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 자일로스, 아라비노스 또는 글루코스 또는 자일로스, 아라비노스 또는 글루코스의 근원을 포함하는 배지에서 숙주 세포를 배양하는 단계를 포함하되, 상기 세포 내로 자일로스, 아라비노스 또는 글루코스의 수송은 재조합 폴리뉴클레오티드의 발현 상에서 증가되었다.
세포 내로 자일로스, 아라비노스 또는 글루코스의 수송은, 실시예 11 에 기재된 방법을 포함하는 본 분야의 숙련자에게 공지된 어떠한 방법으로 측정될 수 있다. 일반적으로 글루코스의 근원은 셀룰로스이고, 자일로스 및 아라비노스의 근원은 헤미셀룰로스이다.
셀룰로스
분해(
degrading
) 방법
본 발명의 하나의 양상은 셀룰로스 분해 방법을 제공한다. 본 방법은 셀룰로스를 포함하는 조성물을 준비하는 첫 번째 단계를 포함한다. 셀룰로스는 바람직하게, 지팽이풀(switchgrass), 미스칸터스 ( Miscanthus ), 왕겨(rice hull), 버개스(bagasse), 아마(flax), 대나무(bamboo), 사이잘(sisal), 마닐라삼섬유(abaca), 짚(straw), 잎(leaves), 잔디 부스러기(grass clippings), 옥수수대(corn stover), 옥수수 속대(corn cobs), 증류립(distillers grains), 콩과 식물(legume plants), 수수(sorghum), 사탕수수(sugar cane), 사탕무 펄프(sugar beet pulp), 우드칩(wood chips), 톱밥(sawdust) 및 바이오매스 크롭(biomass crops)과 같은 식물물지로부터 온 것이다.
본 방법은 변경된 유기체로부터 셀룰라아제-함유 효소 혼합물로부터 조성물을 접촉하는 두 번째 단계를 포함하되, 상기 셀룰라아제-함유 효소 혼합물은, 변경되지 않는 유기체로부터 셀룰라아제-함유 혼합물과 비교하여 감소된 β-글루코시다아제 활성도를 갖는다. 셀룰로스는 셀룰라아제-함유 혼합물에 의해 분해된다. 유기체는 β-글루코시다아제를 코딩하는 유전자 또는 RNA 간섭(RNA interference)과 같은 기술로 β-글루코시다아제의 발현을 감소시키는 유전자의 돌연변이에 의해 변경될 수 있다. 유기체는 균류 또는 세균(bacterium)일 수 있다. 바람직한 실시형태에서, 유기체는 T. 레세이와 같은 곰팡이(filamentous fungus)이다.
선택적으로, 방법은 이의 β-글루코시다아제 활성도를 감소시키기 위해 변경된, 셀룰라아제-함유 효소 혼합물을 갖는 조성물과 접촉하는 두 번째 단계를 포함한다. 예를 들어, 셀룰라아제-함유 효소 혼합물은 친화성 크로마토그래피로 변경될 수 있고, 상기 β-글루코시다아제 효소는 크로마토그래피 동안에 잡힐 수 있고(captured), 이에 의해 혼합물로부터 제거되었다. 또 다른 예에서, 셀룰라아제-함유 효소 혼합물은 저해제(inhibitor)와 함께 혼합물에서 β-글루코시다아제 효소의 불활성화에 의해 변경되었다. 상업적으로 입수가능한 셀룰라아제 혼합물의 예는 Celluclast 1.5L®(Novozymes), Spezyme CP®(Genencor)(Scott W. Pryor, 2010, Appl Biochem Biotechnol) 및 Cellulite 50L(Lyven)이다.
본 발명은 이의 바람직한 특정한 실시형태와 함께 기재된 반면에, 앞서 말한 상세한 설명은 설명하려는 의도일 뿐, 이로 본 발명의 범위를 제한하려는 의도는 아님을 이해할 수 있을 것이다. 본 발명의 범위 내의 또 다른 양상, 이점(advantages) 및 변형은, 본 발명에 적용됨을 본 분야의 숙련자는 명백히 알 수 있을 것이다.
기재된 본 발명 및 하기의 실시예는 설명을 통해 본 발명의 주제를 설명하기 위해 제시하였을 뿐, 이로 제한하려는 의도는 아니다.
미스칸타스
(
Miscanthus
) 및
아비셀(Avicel)에서
성장한
엔. 크라사(N. crassa)
의
전사체
해석(
transcriptome
analysis
)
본 실시예에서, N. 크라사 게놈(genome)의 발현 프로파일(expression profile)은 억세 또는 아비셀에서 성장하는 동안 조사하였다. 유일 탄소원(sole carbon source)은 T. 레세이(T. reesei)(QM9414)(도 3)과 유사한 것으로서, 엔. 크라사(FGSC 2489)의 성장 및 셀룰라아제 활성도(cellulase activity)를, 결정형 셀룰로스(crystalline cellulose)(Avicel로부터 입수)를 갖는 Vogel's 최소배지(minimal media)에서 배양하였다 ; N. 크라사는 대략 4 일에 완전하게 아비셀을 분해하였다. N. 크라사는, 기능적인 셀룰라아제 및 헤미셀룰라아제 변성 능력(degradative capacity)을 제안하는, 그라운드 미스칸타스 줄기(ground miscanthus stem)에서 빠르게 또한 성장하였다. N. 크라사에서 식물 세포 벽 해체(deconstruction)와 관련된 전사체(transcriptome)를 측정하기 위해, 우리는 완전한 게놈 마이크로어레이(full genome microarrays)를 사용하여(Kasuga and Glass 2008; Tian 등의, 2007; Kasuga 등의, 2005), 그라운드 미스칸타스 줄기에서 N. 크라사의 성장 동안에 유전자 발현을 모니터하였다. RNA 는 수크로스에서 16 시간 성장한 후에 표본 조사하였고(sample), 16 시간, 40 시간, 5 일 및 10 일에 미스칸타스 배지에서 성장한 N. 크라사로부터 분리된 RNA 와 비교하였다(도 4 ; Supplemental Data, Dataset SI, page 1 in Tian 등의, 2009 또한 참조 ; bioinfo.townsend.yale.edu/browse.jsp, Experiment IDs 52 and 53 에서 또한 발견할 수 있음).
총 769 N. 크라사 유전자는, 수크로스 샘플과 비교한 바와 같이 4 개의 미스칸터스 샘플 중에서 상대적인 발현 수준에서 통계학적으로 중요한 차이점을 나타내었다(Supplemental Data, Dataset SI, page 3 in Tian 등의, 2009 참조). 계층적 군집(Hierarchical clustering)은 이러한 유전자가 세 가지 주요한 군집(three main clusters) 내에 속함을 나타낸 것이다(도 4 의 A). 유전자의 첫 번째 군집(CI; 300 유전자)은 탄소원(carbon source)으로서의 수크로스를 갖는 최소 배지에서 가장 높은 발현 수준을 나타낸 것이다. 이러한 유전자는 기능적인 카테고리 분석[Functional category(FunCat) analysis](Ruepp 2004)은, 호흡(respiration), 전자 전달 및 아미노산 대사와 같은 일차 물질대사(primary metabolism)와 연관된 리보솜단백질(ribosomal proteins) 및 그 밖의 기능적인 카테로리(functional categories)가 풍부함을 나타낸다(Supplemental Data, Dataset SI, page 4 in Tian 등의, 2009 참조). 두 번째 군집(C2)는 나중 시점(40 시간 내지 10 일)에서 미스칸터스 배양물에서 가장 높은 발현 수준을 나타내는 327 유전자를 포함한다(도 4 의 A). 이러한 그룹 내에서, 모든 시점에서 미스칸터스 배양물에서의 높은 상대적인 발현 수준을 나타낸 것은 89 유전자이다. 추가적인 분석에서, 이러한 89 유전자는 16 시간 미스칸터스 배양물로부터 가장 높은 발현 수준을 나타낸 유전자의 군집에 첨가하였다(C3 군집, 하기를 참조). 하나의 기능적인 카테고리(functional category)(C-화합물 및 탄수화물 대사)에서 나타낸 남아있는 238 유전자의 FunCat 분석(Ruepp 2004)은 약간 강화되었다(enriched)(Supplemental Data, Dataset SI, page 5 in Tian 등의, 2009 참조).
142 유전자의 세 번째 군집은 미스칸터스에서 N. 크라사의 16 시간 성장 후에 상대적으로 가장 높은 발현을 나타내었다(C3, 도 4 의 A). 모든 시점에서 미스칸터스 배양물에서의 높은 발현 수준을 나타낸 이러한 142 유전자 뿐만 아니라 89 유전자의 FunCat 분석(Ruepp 2004)(C3+ 군집 ; 총 231 유전자)은, 예측된 셀룰라아제 및 헤미셀룰라아제를 포함하는 탄소 대사와 연관된 단백질이 풍부함을 나타낸다(도 4 의 C ; 또한 Supplemental Data, Dataset SI, page 6 in Tian 등의, 2009 참조). N. 크라사 게놈에서 23 개의 예측된 셀룰라아제 유전자 중의 18 개는 미스칸터스에서 성장하는 동안, 특히 16 시간 시점에서 발현 수준이 현저한 증가를 나타내었다(도 5)(표 1 in Tian et al., 2009 참조). 5 개의 유전자는 200-폴드 상에서 발현 수준이 증가됨을 나타내었다[cbh-1(CBH(I); NCU07340, gh6-2(CBH(II)-유사 유전자 ; NCU09680), gh6-3(NCU07190) 및 두 개의 GH61 유전자(gh61-4 ; NCU01050 및 NCU07898)].
식물 세포 벽은 셀룰로스 미세섬유, 헤미셀룰로스, 리그린(lignin), 펙틴(pectin), 각피질(cutin) 및 단백질로 이루어진 복합 구조물(complex structures)이다. 따라서, 우리는 미스칸타스에서 성장한 N. 크라사의 발현 프로파일과 아비셀에서 성장한 N. 크라사의 발현 프로파일, 순수한 형태(pure form)의 결정형 셀룰로스(crystalline cellulose)을 비교하였다(Tian 등의, 2009, Supplemental Data, Dataset S I, page 2 를 참조 ; data can also be found at bioinfo.townsend.yale.edu/browse.jsp, Experiment IDs 52 and 53 에서 또한 데이터를 발견할 수 있음). 187 이상 유전자는 아비셀에서 N. 크라사의 성장 동안에 상대적인 발현 수준에서 현저한 증가를 나타내었다. 이러한 유전자 중에 114 는 C3+ 군집에서 231 유전자가 중복되었다(도 4 의 B 참조). 114-오버랩 유전자 세트(the 114-overlap gene set)의 FunCat 분석은 탄소대사에서 포함될 수 있는 것으로 예측된 유전자에 대해서 분명한 풍부함을 나타내었다(Supplemental Data, Dataset S I, page 6 in Tian et al., PNAS, 2009 를 참조). 이러한 유전자 세트내에, 분비된 단백질에 대한 추가적인 풍부함도 있었다 ; 114 개의 유전자 산물 중의 53 개 유전자 산물은 분비될 것으로 예측되었다. 이러한 53 개의 유전자 중에서 32 개의 유전자는 식물 세포 벽 분해(degradation)에서 역할을 제시하는 아노테이션(annotation)을 갖는 것으로 예측된 단백질을 코드하는 반면에 16 유전자는 어떠한 기능적인 예측이 결핍된 것으로 추정되거나(putative) 또는 가상적인 단백질(hypothetical proteins)를 코드한다. 남아있는 61 개의 유전자는 주요한 촉진자 상과 운반체로 예측된 10 개(NCU00801, NCU00988, NCU01231, NCU04963, NCU05519, NCU05853, NCU05897, NCU06138, NCU08114 및 NCU 10021) 및 23 개의 추정되거나 또는 가상적인 단백질을 포함하는, 예측된 세포 내의 단백질을 코드한다.
미스칸터스-특이적인 군집(Miscanthus-specific cluster) 내의 117 개의 유전자 중에 37 개(도 4의 B)는 분비될 것으로 예측되는 단백질을 코드한다. 헤미셀룰로스의 저하(degradation)와 관련된 9 개의 예측된 헤미셀룰라아제 또는 효소는 확인되었다(NCU00710, NCU04265, NCU04870, NCU05751, NCU05965, NCU09170, NCU09775, NCU09923 및 NCU09976)(Tian 등의, 2009 - 표 2). 남아있는 80 개의 미스칸터스-특이적인 유전자는, 오탄당(예를 들어, NCU00891, 자일리톨 탈수소효소 및 NCU00643, 예측된 아라비니톨 탈수소효소)의 대사에 포함되는 유전자를 포함하는 예측된 세포내 단백질, 예측된 당 운반체(NCU01132) 및 알려지지 않은 기능(unknown function)의 48 개의 단백질을 코드한다.
실시예
2 -
미스칸터스
및
아비셀에서
성장한
N.
크라사
의
세크레톰
분석(
Secretome
Analysis
)
진균(fungi)에 의한 리그노셀룰로스 저하(Lignocellulose degradation)는 세포 외에서 일어나고 세포 벽 구성성분(cell wall constituents)의 디폴리머라이제이션(depolymerization)과 연관된 단백질 분비를 필요로 한다(Lynd 등의, 2002). N. 크라사가 미스칸터스 또는 아비셀에서 성장하였을 때, 유전자 코딩하는 것을 예측된 셀룰라아제, 헤미셀룰라아제 및 기타 분비된 단백질은 발현 수준이 증가됨을 나타낸 전사 프로파일링 데이터(transcriptional profiling data)와 비교하기 위해, 우리는 샷건 프로테이믹스 처리 방법(shotgun proteomics approach)을 사용한 N. 크라사의 세크레톰(secretome)을 분석하였다(도 4 의 B). 7 일 된 미스칸터스 및 아비셀 배양물로부터의 상청액을 트립신으로 소화시키고, 액체 크로마토그래피 나노-전자분사 이온화 이중 질량분석기(nano-electrospray ionization tandem mass spectrometry)(MS ; 실시예 5 참조)에 의해 분석하였다. 인산 스웰런 셀룰로스(phosphoric acid swollen cellulose, PASC)에 결합하는 분비된 단백질은 풍부하고(enriched) 또한 MS 에 의해 분석하였다.
총 50 개의 단백질은 이중 MS 에 의해 확실하게 확인할 수 있었다(표 2 및 표 3). N. 크라사 배양물에서 성장한 미스칸터스에서 34 개의 단백질이 발견된 반면에, 배양물에서 성장한 아비셀로부터 38 개의 단백질이 확인되었다 ; 22 개의 단백질은 둘 다의 샘플에서 발견되었다. 이러한 22 개의 단백질 중에, 21 개는 컴퓨터 분석(computational analyses)을 기반으로 분비되는 것을 예측되었고, 19 개는 배양물에서 성장한 미스칸터스 및 아비셀 둘 다에서 발현 수준이 증가됨을 나타내었다(표 2). 오버랩 데이터세트(overlap dataset)는 N. 크라사에서 23 개의 예측된(predicted) 셀룰라아제의 8 개를 포함한다(표 3). 5 개의 예측된 헤미셀룰라아제, 예측된 P-글루코시다아제(gh3 -4; NCU04952), 탄수화물에서 예측된 활성도를 갖는 5 개의 단백질 및 알려지지 않는 기능을 갖는 2 개의 단백질(NCU07143 and NCU05137)이 또한 있었다(표 4-5).
표 2 에 대해서, 아노테이션(annotation)은 웹사이트 broad.mit.edu/ annotation/genome/neurospora/Home.html 에서의 Broad Institute에 의해 생성되었다. "발견된 샘플(sample detected)"은 특정한 단백질에 대해 발견된 펩티드에서의 샘플이다. 펩티드는 점검 매뉴얼(manual inspection)에 의해 승인되었다. 만약 적어도 1 개의 펩티드가 각각의 생물학적 반복(biological repeat)에서 발견된다면 단백질은 존재하는 것으로 결정되었다. "전체(TOTAL)"은 모든 세포외 단백질의 트립신 소화로부터 발견된 펩티드를 언급한다. "PASC BOUND" 는 인산 스웰런 셀룰로스(phosphoric acid swollen cellulose, PASC)에 결합한 단백질이 풍부하게 된(enrichment) 후에 발견된 펩티드를 언급한다. "언바운드(UNBOUND)" 은 PASC 결합 단백질이 제거된 후의 용액에 남아있는 단백질을 언급한다.
[표 2]
[표 3]
[표 4]
[표 5]
아비셀 배양물에서만 16 개의 단백질이 확실하게 확인되었고, 이들 중 14 개는, 두 개의 예측된 셀룰라아제( gh61 -l; NCU02240 및 gh45 -l; NCU05121), 하나의 자일라나아제(xylanase)(ghll -1; NCU02855), 하나의 예측된 프로테아제(protease)(NCU04205), 탄수화물에서 예측된 활성을 갖는 다른 세 개의 단백질[NCU08909, NCU05974 및 gh30 -l (NCU04395)], 알려지지 않는 기능의 세 개의 네우로스포라-특이적인 단백질, 및 셀롤로스 결합 도메인을 갖는 하나의 단백질을 포함하는 4 개의 보존 가설단백질(conserved hypothetical proteins)(NCU09764)을 포함하고 이들은 분비되는 것으로 예측되었다(표 6). 12 개의 단백질은 미스칸터스 배양물의 배양 여과액에 대해 특이적이고, 이들 중 7개는 분비되는 것으로 예측된다(표 3). 5 개의 예측된 세포 내 단백질 중의 3 개는 보전 가설단백질이다. 남은 2 개의 단백질은 예측된 글리옥살 산화효소(glyoxal oxidase)[NCU09267, N. 크 라사 미스칸터스 전사체(transcriptome)로부터 확인됨] 및 뉴클레오시드 이인산염 키나아제(nucleoside diphosphate kinase)[ndk -1; NCU04202, N. 크라사 미스칸터 스 전사체로부터 확인되지 않음]를 포함한다. 7 개의 단백질은, 3 개의 예측된 에스테라아제(NCU04870, NCU05159 및 NCU08785), 두 개의 예측된 자일라나아제(GH51 ; NCU02343 및 GH54 ; NCU09775), 예측된 P-자일로시다아제(gh3 -7; NCU09923) 및 보존 가설단백질(NCU05751)을 포함하고, 분비되는 것으로 예측되었다.
[표 6]
많은 식물 세포벽 분해효소(plant cell wall degrading enzymes)는, 기질에 효소를 부가하는 셀룰로스-결합 모듈(cellulose-binding module, CBM)을 함유한다(Linder and Teeri 1996). N. 크라사 게놈 내의, 19 가지 유전자를 코드하는 단백질은 CBM1 도메인을 함유하는 것을 예상하였다(Cantarel 등의, 2009). 이러한 19 가지 유전자 중에, 16 가지는 미스칸터스-성장한 배양물에서 상대적인 유전자 발현에 있어서 증가됨을 나타내었다(표 7).
[표 7]
MS 에 의해 확인된 50 개의 단백질로부터, 11 개는 CBM1 도메인을 함유한다. PASC 는 셀룰로스에 결합하는 단백질을 풍부하게 하기 위해(enrich) 사용되었다(방법에 대해서 실시예 4 에 참조). 9 개의 단백질은 N. 크라사 배양물에서 성장한 미스칸터스의 상청액으로부터의 PASC 에 결합하는 MS 에 의해 확인한 반면에, 아비셀 상청액으로부터의 8 개의 단백질을 확인하였다 ; 7 개의 셀룰로스 결합 단백질은 둘 다에서 확인되었다(표 2, 3 및 8). 이들은 NCU00206, 예측 셀로비오스 탈수소효소(cellobiose dehydrogenase) ; gh5 -l(NCU00762), 예측 엔도글루카나아제(predicted endoglucanase) ; NCU05955, 예측 GH74 자일로글루카나아제(xyloglucanase) ; ghll -2(NCU07225), 예측 엔도자일라나아제(predicted endoxylanase) ; cbh -1(NCU07340) ; gh61 -5(NCU08760), 예측 엔도글루카나아제 ; 및 gh6 -2(NCU09680), 예측 셀로비오히드롤라아제 2 전구체(predicted cellobiohydrolase 2 precursor)를 포함한다.
[표 8]
전사체
/
세크레톰
데이터세트(
Transcriptome
/
Secretome
Datasets
)의 오버랩(
Overlap
)에서 확인된 유전자에서의 결실(
Deletions
)을 함유하는 스트레인에서 세포외 단백질(
Extracellular
Proteins
) 및 셀룰라아제 활성도(
Cellulase
Activity)의 특성
배양물에서 성장한 미스칸터스 및 아비셀 둘 다에서 관찰된 22 가지의 세포외 단백질 중에서, 이들 세포외 단백질 중 16 가지를 코딩하는 유전자에서 결실을 함유하는 동종핵형(homokaryotic) 스트레인은 일반적으로 입수할 수 있다(Dunlap 등의, 2007). 이들 16 가지의 결실 스트레인은 N. 크라사에서 식물세포벽 또는 셀룰로스 분해에 서 이들의 영향에 대해서 이전에 특징지어지지 않았다. 16 가지의 결실 스트레인은 바람직한 탄소원(carbon source)으로서 수크로스 또는 아비셀을 함유하는 배지에서 모두 성장하였다. 모든 스트레인은 수크로스에서 야생형 성장 형질형(wild type growth phenotype)을 나타내었다. 아비셀을 함유하는 배지에서, 16 가지 결실 스트레인의 대부분의 성장을 7 일 동안 모니터하였다. 7 일 후에, 총 분비된 단백질(secreted protein), 엔도글루카나아제 활성도(endoglucanase activity), β-글루코시다아제 활성도 및 배양 여과액(culture filtrates)의 총 아비셀라아제 활성도(aggregate Avicelase activity)를 측정하였고, 유도된 모든 돌연변이체(mutants)로부터의 야생형 스트레인과 비교하였다(도 6). SDS-PAGE 는 분비된 단백질의 상대 빈도(relative abundance)를 조사하기 위해 농축시키지 않은 배양 상청액(unconcentrated culture supernatants)에서 또한 실시하였다.
두 가지의 예측 엑소글루카나아제(cbh -1 ; NCU07340 및 gh6 -2 ; NCU09680) 및 예측 β-글루코시다아제(gh3-4 ; NCU04952)의 결실을 포함하는 스트레인에 대해서 아비셀에서의 성장 결핍(growth deficiencies)이 있었다. cbh -1 돌연변이체는 가장 심각하다 ; 7 일 후에 대부분의 아비셀은 남아있는 반면에, 야생형 스트레인의 모든 아비셀은 이 시점에 분해되었다. 16 개의 결실 스트레인 중 10 개에 대해서, 분비된 단백질의 SDS-PAGE 분석은 단일 밴드가 사라진 곳에서 변화된 세포외 단백질 프로파일(altered extracellular protein profile)을 나타내었고, 따라서 예측 유전자에 대한 특정 단백질 밴드(protein band)의 배치(assignment)를 가능하게 한다(도 6A, 박스(boxes) ; 도 7). 이들은 NCU00762( gh5 -l), NCU04952( gh3 -4), NCU05057( gh7 -l), NCU05137, NCU05924(ghlO -1), NCU05955, NCU07190( gh6 -3), NCU07326, NCU07340(cbh -1) 및NCU09680( gh6 -2)을 포함한다.
결실 스트레인의 대부분에 대해서, 전체 분비된 단백질, 엔도글루카나아제, β-글루코시다아제 및 배양 상청액의 아비셀라아제 활성도(Avicelase activities)는 야생형(wild type)과 유사하였다(도 6B, C 및 표 9).
[표 9]
이러한 경향(trend)으로부터의 편차(Deviations)는 △gh5 -l(NCU00762), △gh3-4(NCU04952), △NCU05137, △cbh -1(NCU07340), 및 △gh6 -2(NCU09680) 돌연변이체(mutants)에서 볼 수 있다. △gh5 -l (NCU00762), △gh3 -4 (NCU04952) 및 △cbh -1 (NCU07340)에서, 아비셀라아제(Avicelase), 엔도글루카나아제(endoglucanase) 또는 △-글루코시다아제 활성도는 야생형의 활성도에 상응하는 것 보다 낮았다. 특히, NCU04952의 결실(deletion)은, 아비셀라아제 효소 검정(Avicelase enzyme assays)에서 PNPGase 활성도 및 높은 수준의 셀로비오스 및 낮은 수준의 글루코스에 의해 입증된 바와 같이, 배양 상청액으로부터의 모든 β-글루코시다아제 활성도를 제거하였다(도 6B 및 C). 낮은 엔도글루카나아제 활성도에도 불구하고, △gh5 -l (NCU00762)로부터의 배양 여과액(culture filtrate)은 야생형 스트레인에 관하여 아비셀룰라아제 활성도에서 현저한 결핍을 나타내지 않았다(도 6C). 예상대로, cbh-1(NCU07340)에서의 돌연변이는 좋지 못한 성장 때문에, 엔도글루카나아제 및 아비셀룰라아제 활성도가 더 낮아졌다. CBH(II)-유사 단백질(gh6 -2)을 코딩하는 NCU09680 의 결실을 포함하는 스트레인은 △cbh -1 돌연변이체(mutant)에서 관찰된 바와 같이, 감소된 셀로비오스 축적(accumulation)을 또한 나타내었다(도 6 C).
세 가지의 스트레인에서 돌연변이로 인하여 특히 CBH(I)(도 6A); gh3 -4 (NCU04952), gh7 -l(NCU05057) 및 가설단백질 유전자(hypothetical protein gene) (NCU05137)의 수준이 증가되었다. 분비된 단백질의 수준이 증가될 뿐 아니라, △NCU05137 돌연변이체는 증가된 엔도글루카나아제, β-글루코시다아제 및 아비셀룰라아제 활성도가 증가됨을 나타내었다(도 6 B 및 C). NCU05137 은 그 밖의 섬유소 분해성 균류(cellulolytic fungi)을 포함하는 수많은 곰팡이 자낭기(filamentous ascomycete fungi)의 게놈에서 매우 잘 보존되어 있지만 T. 레세이( T. reesei )에는 현저히 오솔로그(ortholog)를 갖지 않았다(도 2). 증가된 분비, 단백질 안정성 또는 그렇지 않으면 cbh - 1 의 발현에서의 증가를 결과로 나타내는 피드백으로 인하여 △gh3-4, △ gh7 -1, 및 △NCU05137 에서 관찰된 CBH(I) 수준이 증가될 수 있다. 이러한 가능성의 구별을 위해서, 정량적 RT-PCR 에 의한 검정에 의하면, △NCU05137 및 △gh3 -4(NCU04952)에 의해 생산된 세포외 단백질(extracellular proteins)의 프로파일(profile)은 cbh -1(NCU07340) 및 gh6 -2(CBH(II); NCU09680)의 유전자 발현 수준과 비교하였다(도 8). 스트레인 △NCU05137 및 △gh3-4 은 아비셀-성장 배양물에서 이미 2 일에 CBH(I) 단백질의 수준이 더 높아졌다. 둘 다의 유전자가 성장 2 일 후에 야생형 및 △NCU05137 및 △gh3 -4 돌연변이체에서 높은 발현 수준을 나타냄을, 아비셀-성장 배양물로부터의 cbh -1 및 gh6 - 2 의 정량적 RT-PCR 에서 나타내었다. 그러나, 이러한 유전자 모두의 발현이 야생형 스트레인에서 3 일째에 현저하게 감소되었음에도 불구하고, cbh -1 및 gh6 -2 둘 다에서 발현 수준은 △NCU05137 돌연변이체에서 증가되었고, △gh3 -4 에 야생형보다 덜 감소되었다(도 8). △NCU05137 및 △gh3 -4 돌연변이체에서의 cbh -1 and gh6 -2 유전자의 지속적인 발현(Sustained expression)은 CBH(I) 및 CBH(I) 단백질 수준에서 관찰된 증가에 대한 책임이 있을 수 있다.
전사체
(
Transcriptome
) 및
세크레톰
(
Secretome
) 연구에 대한 물질 및 방법
스트레인
Fungal Genetics Stock Center 로부터 모든 네우로스포라 크라사 스트레인을 입수하였다(FGSC; 웹페이지 fgsc.net)(Supplemental Data, Dataset SI, page 1 in Tian 등의, 2009). 유전자 결실 스트레인은 N. 크라사 기능 유전학 프로젝트(functional genomics project)로부터 입수하였다(Dunlap 등의, 2007). 트리코데르마 레세이 ( Trichoderma ressi ) QM9414 은 Dr. Monika Schmoll로부터의 선물이다(비엔나 기술공과대학). 2 %(w/v) 탄소원을 갖는 Vogel's 염에서 스트레인을 성장시켰다[미스칸터스, 수크로스 또는 아비셀(Sigma)]. 미스칸터스 x 기간테우스( Miscanthus x giganteus )[-0.1 mm 에 대한 밀드 스템(milled stem)]은 일리노이노 대학으로부터의 선물이다.
효소 활성도 측정
Bio-Rad DC 단백질 분석 키트[Bio-Rad DC Protein Assay kit(Bio-Rad)]를 사용하여 전체 세포외 단백질 함량을 측정하였다. 배양 상청액에서 엔도글루카나아제 활성도를 azo-CMC 키트(Megazyme SCMCL)로 측정하였다. 10 분 동안 40 ℃ 에서, pH 5.0 의 50 mM 아세트산 나트륨 완충용액에서 500 μM 4-니트로페닐 β-D-글루코피라노시드(4-nitrophenyl P-D-glucopyranoside)와 10 배 희석한 배양 상청액(10-fold diluted culture supernatant)을 혼합하여 β-글리코시다아제 활성도를 측정하였다. 반응물을 5 % w/v 탄산나트륨으로 퀀칭하였고(quenched), 400 nm 에서 흡광도(absorbance)측정하였다. 40 ℃ 에서 50 mM 아세트산나트륨 및 5 mg/mL 의 아비셀과 2 배 희석한 배양 상청액을 혼합하여 아비셀라아제 활성도를 측정하였다. 상청액(Supernatants)을, 글루코스 산화효소/페록시다아제(glucose oxidase/peroxidase)로 결합된 효소 검정(coupled enzyme assay)을 사용하여 글루코스 함량(glucose content)에 대해 분석하였다. 50 μL 의 아비셀라아제 반응물(avicelase reaction)을, pH 5.0 의 100 mM 의 아세트산 나트륨, 10 U/mL 의 겨자무과산화효소(horseradish peroxidase), 10 U/mL 의 글루코스 산화효소 및 1 mM o-디아니시딘(o-dianisidine)을 함유하는 150 μL 의 글루코스 검출시약(glucose detection reagent)에 이동시켰다. 30 분 후에, 540 nm 에서 흡수(absorption)를 측정하였다. 셀로비오스 농도는 스포로트리쿰 서모필( Sporotrichum thermophil)로부터의 셀로비오스 탈수소효소(cellobiose dehydrogenase, CDH)와 함께 결합된 효소 검정(coupled enzyme assay)을 사용하여 측정하였다. CDH 는 이전의 보고와 유사한 S. 서모필로부터 분리하였다(isolate)(Canevascini 1988). 50 μL 의 아비셀라아제 반응물을, pH 5.0 의 125 mM 아세트산 나트륨, 250 μM 디클로로페놀 인도페놀(dichlorophenol indophenol) 및 0.03 mg/mL CDH 을 함유하는 250 μL 의 셀로비오스 검출시약(cellobiose detection reagent)에 이동시켰다. 10 분 후에, 530 nm 에서 흡수(absorption)를 측정하였다.
RNA
분리,
마이크로어레이
분석(
Microarray
Analysis
) 및 신호 펩티드 예측
균사체(Mycelia)를 여과 및 액체질소에서 냉동시킨 플레시(flash frozen)로 채취하였다(harvested). 전체 RNA 를 트리졸(trizol)을 사용하여 분리하였다(Tian 등의 , 2007 ; Kasuga 등의., 2005). 마이크로어레이 혼성(Microarray hybridization) 및 데이터 분석을 상기한 바와 같이 하였다(Tian 등의, 2007). 정규화된 발현값을, 각각의 실험 시점에서 각각의 유전자에 대한 상대적인 유전자 발현 수준(relative gene expression levels) 및 믿을 수 있는 인터벌(credible interval)을 추론할 수 있는 BAGEL[유전자 발현 수준의 베이지안 분석(Bayesian analysis of gene expression levels)]을 사용하여 분석하였다(Townsend 2004; Townsend and Hartl 2002). 신호펩티드는 신호P3 프로그램(signalP3 program)을 갖는 각각의 예측 단백질의 N-터미널(terminal) 70 개의 아미노산 영역을 사용하여 예측할 수 있다(웹페이지 cbs.dtu.dk/services/SignalP-3.0/). 원본 프로파일링 데이터를 (웹페이지 yale.edu/townsend/Links/ffdatabase/)에서 입수하였다.
단백질 겔 전기이동(
Protein
Gel
Electrophoresis
)
다른 방법으로 언급한 경우를 제외하고, 기준 4-15 % 트리스-HCl 폴리아크릴아미드 겔(Criterion 4-15% Tris-HCl polyacrylamide gels) 내로 로딩하기 전에, 농축시키지 않은 배양 상청액을 5x SDS 로딩 염료(loading dye)로 처리하고, 5 분 동안 끓였다(boiled). 쿠마시 염료(Coomassie dye)를 염색(staining)에 사용하였다.
세크레톰
분석에 대한 트립신 분해 펩티드(
Tryptic
Peptides
)의 제조
배양 상청액을 10 kDa MWCO PES 스핀 농축기(spin concentrators)로 농축시켰다. 셀룰로스 결합 단백질을 인산 스웰런 셀룰로스(PASC)의 첨가에 의해 배양 상청액으로부터 분리하였다. 10 mg/mL PASC 의 5 mL 의 현탁액(suspension)을 10 mL 의 배양 상청액에 첨가하였다. 5 분 동안 4 ℃ 에서 배양한 후에, 혼합물을 원심분리하고 펠렛된(pelleted) PASC 를 pH 5.0 의 100 mM 아세트산나트륨의 20 펠렛 부피(pellet volumes)로 세척하였다. PASC 로 처리한 후에 상청액을 결합하지 않은 분획으로 저장하고 농축시켰다. 36 mg 의 요소, 5 μL 의 1 M 트리스 PH 8.5 및 5 μL 의 100 mM DTT 를 100 μL 의 농축된 배양 상청액 또는 단백질-결합 PASC 에 첨가하였고, 혼합물을 1 시간 동안 60 ℃ 에서 가열하였다. 700 μL 의 25 mM 중탄산암모늄 및 140 μL 의 메탄올을 상기 용액에 첨가한 다음에 pH 5.0 의 50 mM 아세트산 나트륨에 용해시킨 50 μL 의 100 ㎍/mL 트립신으로 처리하였다. PASC 결합 단백질에 대해서, PASC 를 가열한 후에 원심분리하여 제거한 다음에, 상청액을 트립신으로 처리하였다. 트립신을 37 ℃ 에서 밤새 반응시키도록 두었다. 분해(digestion)시킨 후에, speedvac 으로 부피(volume)를 줄였고, 3 번 MilliQ 물로 세척하였다. 샘플(sample)에서 잔류염(Residual salt)을 제조자의 지시(manufacturer's instructions)에 따라 OMIX 마이크로추출법 피펫팁(OMIX microextraction pipette tips)을 사용하여 제거하였다.
트립신 분해 펩티드(
Tryptic
Peptides
)의 액체 크로마토그래피
트립신-분해 단백질을 고성능 액체크로마토그래피(ultraperformance liquid chromatography, UPLC)가 인-라인(in-line)으로 연결된 이중질량분석기(tandem mass spectrometer)를 사용하여 분석하였다. C18 트래핑(trapping)(180 μm x 20 mm) 및 분석(100 μm x 100 mm) 컬럼 및 10 μL 샘플 루프가 갖추어진 nanoAcquity UPLC(미국 매사추세츠 밀퍼드에 위치한 Waters)을 사용하여 펩티드를 분리하였다. 용매 A 를 0.1 % 의 포름산/99.9 % 의 물이고, 용매 B 는 0.1 % 의 포름산/99.9 % 의 아세토니트릴(v/v)이다. 셉타 캡스(septa caps)로 밀봉된 0.3 mL 의 폴리프로필렌 스냅-톱 유리병(snap-top vials)에 포함된 샘플 용액은 분석 전에 nanoAcquity 오토샘플러(autosampler) 내에 로딩하였다. 샘플 주입[2 μL, 부분적인 루프(partial loop)] 후에, 트래핑(trapping)을 3 μL/분의 유량(flow rate)에서 100 % A 를 가지고 5 분 동안 실시하였다. 샘플 사이에 교차-오염(cross-contamination)을 방지하기 위해 주입 후에 각각의 용액 A 및 B 의 750 μL 로 주입 바늘(injection needle)을 세척하였다. 용출 프로그램은 500 nL/분의 유량(flow rate)으로, 55 분 이상 동안 25 % 에서 30 % B 까지의 선형 구배(linear gradient), 20 분 이상 40 % B 까지의 선형구배, 0.33 분 이상 95 % B 까지의 선형 구배, 11.67 분 동안의 95 % 에서의 등용리 조건(isocratic conditions), 0.33 분 상의 1 % B 까지의 선형 구배 및 11.67 분 동안의 1% B 에서의 등용리 조건으로 구성되었다. 분석 컬럼 및 샘플 컴파트먼트(sample compartment)를 각각 35 ℃ 및 8 ℃ 에서 유지시켰다.
질량 분석법(
Mass
Spectrometry
)
컬럼을 사중극 비행 시간 분석기(quadrupole time-of-flight mass spectrometer)(Q-Tof Premier, Waters)의 나노 유속 이온원(nanoflow ion source)에 설치한 NanoEase 나노일렉트로 스프레인 이온화(nanoESI) 에미터[NanoEase nanoelectro spray ionization (nanoESI) emitter]에 컬럼을 연결하였다. nanoESI 소스 파라미터(source parameters)는 다음과 같다 ; nanoESI 모세관 전압(capillary voltage) 2.3 kV, 분무 가스(nebulizing gas)(질소) 압력 0.15 mbar, 샘플 콘 전압(sample cone voltage) 30 V, 추출액 콘 전압(extraction cone voltage) 5 V, 이온 가이드 전압(ion guide voltage) 3 V, 및 소스 블록 온도(source block temperature) 80 ℃. 어떠한 콘 가스(cone gas)도 사용하지 않았다. 충돌 셀(collision cell)은 8 x 10-3 mbar 의 압력으로 아르곤 가스를 함유한다. Tof 분석기(analyzer)는 "V" 모드(mode)에서 작동하였다. 이러한 조건 하에서, 질량 분해능1(mass resolving powerl)의 1.0 x 104( m/z = 771 에서 측정)은 일상적으로 달성하였고, 이는 이러한 연구에서 측정된 1가 및 다가 펩티드 이온(singly and multiply charged peptide ions)의 동위 원소 분포(isotopic distributions)를 해결하기(resolve) 충분하다. 따라서, 이온의 질량 및 전하(charge)는 독립적으로 결정될 수 있고, 즉 이온 전하는 m/z 스펙트럼에서 인접한 동위원소 피크(adjacent isotope peaks) 사이의 상호간의 간격(reciprocal of the spacing)으로부터 결정된다. 포름산 나트륨(sodium formate)의 용액을 사용하여 외부의 질량 검정(External mass calibration)은 분석 전에 즉시 실시하였다. 조사 스캔(Survey scans)을, 0.95s 스캔 인테그레이션(scan integration) 및 0.05s 인터스캔 지연(interscan delay)을 사용한 범위 m/z = 450-1800 상에서 양성 이온 모드에서 획득하였다. 데이터-의존적인 모드에서, 35 카운트/초(cps)의 강도 한계점(intensity threshold)을 초과한 5 까지의 전구이온(precursor ions)은 이중 질량 분석(MS/MS)에 대한 각각의 조사 스캔(survey scan)으로부터 선택하였다. 리얼-타임 데이소토핑(Real-time deisotoping) 및 전하 상태 인식(charge state recognition)은 MS/MS 에 대한 2+, 3+, 4+, 5+ 및 6+ 전하 상태 전구이온(charge state precursor ions)을 선택하기 위해 사용되었다. 충돌적인 활성화된 해리(collisionally activated dissociation, CAD)에 대한 충돌 에너지(Collision energies)는 주어진 전구이온(precursor ion)의 질량 및 전하 상태를 기반으로 자동적으로 선택된다. MS/MS 스펙트럼은 0.95s 스캔 인테그레이션(scan integration) 및 0.05s 인터스캔 지연(interscan delay)을 사용한 범위 m/z = 50-2500 상에서 획득하였다. 이온을, 최대 3 초 동안 누적(cumulative) MS/MS 스펙트럼에서 30,000 cps 의 최소한의 총 이온전류(ion current)(TIC)를 성취하기 위해 해체하였다(fragment). 불필요한 MS/MS 측정의 발생을 피하기 위해서, 리얼 타임 배제(real time exclusion)는 180 초 동안의 + 0.25 m/z 단위의 배제 너비(exclusion width) 상에서 사전에 분석한 전구이온의 재-선별(re-selection)을 불가능하게 하기 위해 사용되었다.
질량 분석 데이터 분석
트립신-분해 단백질의 LC-MS/MS 분석 결과의 데이터는 ProteinLynx Global Server software (version 2.3, Waters)을 사용하여 처리하였고, 이는 질량 스펙트럼 및 MS/MS 스펙트럼의 배경 차감[한계점(threshold) 35% 및 5차 다항식(fifth order polynomial), 스무딩(smoothing)[Savitzky-Golay2 10 타임즈(times), 세 채널 상에서(over three channels)] 및 센트로딩(centroiding)[반 높이 네 체널(half height four channels)에서의 각각의 피크(peak)의 톱(top) 80 % 및 최소 피크 너비(minimum peak width)]에서 수행하였다. 가공 데이터(processed data)는 하기의 기준(criteria)을 사용하여 N. 크라사 데이터베이스와 비교하여 검색하였다 ; 5 까지의 손실된 분열(missed cleavages)를 갖는 트립신 분해 단편(tryptic fragments), 전구이온 질량 허용오차(precursor ion mass tolerance) 500 ppm, 단편 이온 질량 오차 0.1 Da, 그리고 하기의 가변적인 번역후의 변형물(post-translational modifications) : N-말단의 카르바밀화(carbamylation) 및 Lys 잔기(side chains), Met 산화 및 Ser/Thr 탈수(dehydration). 동일한 시리즈로부터 적어도 세 개의 연이은 단편 이온의 식별, 즉 b 또는 y-타입 단편 이온은 MS/MS 스펙트럼에 대한 펩티드의 배치(assignment)를 요구한다. MS/MS 스펙트럼은 상기 펩티드를 유일하게 확인하기 위한 단편 이온의 존재를 확인하기 위해 매뉴얼적으로(manually) 점검하였다.
정량적인(
Quantitative
)
RP
-
PCR
RT-PCR 을 Qiagen 로부터의 시약으로 AB17300 에서 수행하였다[SYBR-green RT- PCR kit (Cat No. 204243)]. CBHI(NCU07340)에 대한 프라이머(primers)는 포워드(forward) 5'-ATCTGGGAAGCGAACAAAG-3'(SEQ ID NO: 16) 및 리버스(reverse) 5'-TAGCGGTCGTCGGAATAG-3'(SEQ ID NO: 17)이다. CBHII(NCU09680)에 대한 프라이머는 포워드 5'-CCCATCACCACTACTACC- 3'(SEQ ID NO: 18) 및 리버스 5'-CCAGCCCTGAACACCAAG-3'(SEQ ID NO: 19)이다. 정규화(normalization)에 대한 대조군(control)으로서 액틴(Actin)을 사용하였다. 액틴에 대한 프라이머는 포워드 5'-TGA TCT TAC CGA CTA CCT-3'(SEQ ID NO: 20) 및 리버스 5'-CAG AGC TTC TCC TTG ATG -3'(SEQ ID NO: 21)이다. 정량적인 RT-PCR 은 Dementhon 등의(2006)에 따라 수행하였다.
전사체
및
세크레톰
연구의 논의
식물 바이오매스의 분해는 많은 상이한 효소의 활성도의 생산을 요구하고, 이는 이용할 수 있는 식물 물질(available plant material)의 유형(type) 및 복잡성(complexity)에 의해 조절된다(도 9)(Bouws 등의, 2008). 섬유소 분해 균류(cellulolytic fungus)에 의한 식물 세포 벽 분해(plant cell wall degradation)의 첫 번째 체계적인 분석(systematic analyses)은 본원에 기재되어 있고, 이는 전사체(transcriptome), 세크레톰(secretome) 및 돌연변이체(mutant) 분석을 포함한다. 프로파일링 데이터(Profiling data)는, 미스칸터스 또는 아비셀에서의 성장에 의한 도전을 할 때, N. 크라사는 세포외(extracellular) 및 세포내(intracellular) 단백질의 숙주(host)를 대등하게 발현시킴을 나타내었다(도 9). 셀룰로스 기질(cellulosic substrates)에서 성장하는 동안에 가장 많이 발현된 유전자의 대부분은 식물 세포 벽 다당류의 물질대사(metabolism)에 포함되는 것으로 예측된 단백질을 코드하고, 이들 중의 다수는 MS 분석에 의해 확인되었다. 곰팡이(filamentous fungi)의 게놈 비교는 N. 크라사와 밀접하게 덩-분해 종(dung-degrading species), T. 레세이(Martinez 등의., 2008)에서 10 부터 포도스포라 앤서리나(Espagne 등의., 2008)에서 60 까지의 많은 수의 예측된 셀룰라아제와 많은 수의 글리코실 가수분해 효소(glycosyl hydrolases)(-200)를 나타낸다. 백색 부유 담자균(white rot basidiomycete fungus), 파네로케이트 크리소스포리움( Phanerochaete chrysosporium)(Wymelenberg 등의, 2009)에서 최근 전사체/세크레톰 연구(transcriptome/secretome study) 및 이들의 결과 사이의 비교는, 둘 다의 종(species)이 순수한 셀룰로스(pure cellulose)에서 성장하였을 때, 조절된 유전자(regulated genes)(18 유전자) 및 분비된 단백질(2 단백질)에서 약간의 중복(overlap)을 나타내었다. 이러한 데이터는 식물 세포벽 분해를 위한 상이한 유전자 세트(gene sets)를 사용하는 것을 제안한다. 그러나, 연구 모두가 공동으로 갖는 하나의 측면은 셀룰로스 분해(cellulose degradation)와 관련된 많은 수의 특징지어지지 않은(uncharacterized) 유전자/단백질이다. P. 크리소스포륨(P. chrysosporium)을 포함하는 그 밖의 섬유소분해성 균류(cellulolytic fungi)는 N. 크라사와 함께 손쉽게 응용할 수 있는 유전적이고 분자로 구성된 기술(genetic and molecular tools)을 갖지 않는다. N. 크라사와 함께 이용가능한 기능적인 유전적인 기술(functional genomic tools)을 사용하여, 식물 세포벽 분해 효소 시스템의 기능 및 불필요한 반복(redundancy) 둘 다는 리그노셀룰로스 바이오매스(lignocellulose biomass)로부터 액체 연료(liquid fuels)의 산업적인 생산을 위한 최적의 효소 혼합물(optimal enzyme mixtures)을 생산하기 위해 연구할 수 있다.
이러한 연구에서, N. 크라사에서의 셀로비오하이드로라아제(cellobiohydrolase)(I)(CBHI)가 아비셀 또는 미스칸터스에서 성장하는 동안에 가장 많이 생산된 세포외 단백질(extracellular protein)임이 밝혀졌고, 이러한 유전자의 결실은 셀룰로스 기질(cellulosic substrates)에서 가장 심각한 성장 결핍증(deficiencies)을 일으켰다. 이러한 결과는 T. 레세이에서 보고된 결과와 유사하다(Suominen 등의., 1993, Seiboth 등의, 1997). 셀로비오하이드로라아제(II)의 결실은 셀룰로스계 기질(cellulosic substrates)에서의 성장 결핍증을 또한 유발하지만 CBH(I) 보다는 더 적은 정도이고, 이는 N. 크라사에서의 엑소글루카나아제 활성도(exoglucanase activity)는 CBH(I)으로부터가 대부분이고, 셀룰라아제 및 그 밖의 CBHs는 CBH(I)의 손실(loss)에 대해서 보상하지 않는 점을 나타내었다. 여기에서, 셀룰로스계 기질에서 성장하는 동안 가장 많이 생산된 세 가지 엔도글루카나아제는 NCU05057, NCU00762, 및 NCU07190 에 의해 코드됨을 나타내었다. 이러한 단백질은 각각 엔도글루카나아제 EG1, EG2 및 EG6 와 상동(homology)이다. 분비된 단백질 수준 및 엔도글루카나아제 활성도에서의 상이한 점은 관찰되지 않았지만, 이러한 유전자의 결실은 아비셀에서의 성장에 영향을 주지 않는다. 예상외로, △NCU05057 스트레인에서, 세포외 단백질 수준은 특히 CBH(I) 에서 보다 높았고, 이는 결정질 셀룰로스(crystalline cellulose)에서 야생형 성장 표현형(wild- type growth phenotype)을 유지시키기 위해, 돌연변이체(mutant)는 그 밖의 셀룰라아제의 생산을 증가시키기 위해 집중되거나(force), 또는 NCU05057 촉매의 생산은 셀룰라제 생산을 억제할 수 있음을 나타낸다. N. 크라사에서 하나의 엔도글루카나아제는 결정질 셀룰로스에서의 성장을 필요로 하지 않고 상이한 엔도글루카나아제는 중복된 기질 특이성(overlapping substrate specificities)을 갖음을 결론으로 내렸다.
글리코시드 히드로라아제 패밀리(family) 61 효소는 T. 레세이와 비교하여 N. 크라사에서 크게 확대되었다(Martinez 등의, 2008). 이러한 효소는 저조하게 규정된 생물학적 기능(biological function)을 갖지만, 섬유소분해성 균류(cellulolytic fungi)에서의 이들의 일반적인 보존(general conservation) 및 풍부(abundance)는 식물 세포벽 물질대사에서 중요한 역할을 함을 암시한다. 여기에서, 14 가지의 GH61 효소 중의 10 가지에 대한 유전자는 N. 크라사 전사체(transcriptome)에서 확인되었고, 이는 이러한 효소는 셀룰로스계 바이오매스(cellulosic biomass)에서 성장하는 동안 사용됨을 암시한다. 실험된 4 가지 GH61 결실 스트레인은 분비된 단백질 수준, 엔도글루카나아제 및 전체 셀룰라아제 활성도에서 야생형과 비교하여 오직 작은 차이점을 나타내었다. 그러나, 유성교차(sexual crosses)을 통해 N. 크라사에서의 다양한 돌연변이를 포함하는 스트레인을 생산하기 위해 추가적인 GH61 돌연변이체 및 능력의 분석은 불필요한 중복(redundancy)을 다룰 것이고, 이러한 패밀리에서 기능적인 분석(functional analysis)을 더 신속히 처리할 수 있을 것이다.
예측된 셀룰라아제 유전자 뿐 아니라, 탄수화물에서 활성도를 갖는 것으로 예측된 헤미셀룰라아제, 탄수화물 에스테라아제(carbohydrate esterases), β-글루코시다아제, β-자일로시다아제(xylosidases) 및 그 밖의 단백질은 미스칸터 스 및 아비셀 둘 다로부터 N. 크라사 전사체에서 확인되었다. 아비실이 헤미셀룰로스 성분을 함유하지 않다는 사실은, 셀룰로스가 아마도 N. 크라사에서 식물 세포벽 분해 효소를 코딩하는 유전자의 주된 유도물질(primary inducer)임을 암시한다. 그러나, 몇몇의 헤미셀룰라아제 및 탄수화물 에스테라아제를 코딩하는 유전자는 미스칸터스에서 성장하는 동안만 발현되었다. T. 레세이 및 아스페르길루스 니가( Aspergillus niger )와 같은 그 밖의 섬유소분해성 균주에서 유사하게, 몇몇의 셀룰라아제 및 헤미셀룰라아제를 코딩하는(encoding) 유전자는 대등하게 조절된 반면에, 그 밖에는 별도로 조절되었다(Strieker 등의., 2008). 예상대로, NCU05137 및 gh3-4 을 제외하고, 비-셀룰라아제 유전자의 삭제는 아비셀 또는 셀룰라아제 활성도에서의 성장에 작은 영향을 준다. 보다 많은 단백질을 분비하는 △NCU05137 스트레인은 보다 높은 셀룰라아제 활성도를 갖고, 이는 야생형보다 cbh -1(CBH(I)) 및 gh6 -2 (CBH(II))의 보다 높은 발현을 나타내었다. NCU05137 는 알려진 기능(known function)의 단백질과 상동적이지 않은 분비된 가설단백질(secreted hypothetical protein)을 코드하지만, 다른 섬유소분해성 균류에서 매우 잘 보존되었다(도 2 ㅣ E 값 0.0). NCU05137 은 보다 거리가 있는 상동성(more distant homologs)을 갖지만, 수많은 균종(bacterial species)에서 알려지지 않은 기능 또한 갖는다. NCU05137 의 단백질 산물은 셀룰라제 유전자 발현 N. 크라사의 유도와 관련된 신호전달 과정(signaling processes)에 개입할 수 있다(도 9). 유사하게, gh3 -4(NCU04952)에서의 돌연변이는 CBH(I) 활성도를 또한 증가시킨다. 이러한 유전자의 결실은 PNPGase 활성도를 완전하게 제거하고, 셀로비오스는, △gh3-4 배양 여과액(culture filtrates)을 사용한 생체외 ( in vitro ) 셀룰라아제 검정에서 축적되었다. 모든 데이터는, NCU04952 가 N. 크라사에서 주된 세포외 β-글루코시다아제를 코드함을 제시한다. 이러한 데이터는 글루코스에 의한 셀룰라아제 생산의 분해대사물억제(catabolite repression)와 일치한다.
셀룰로스 및 헤미셀룰로스의 세포외 분해(Extracellular degradation)로 세포 내로 순차적으로 수송되는 가용성 탄수화물(soluble carbohydrates)이 형성되었다(도 9). 이러한 연구에서, 투과효소(permeases)/운반체(transporters)를 코드하는 10 가지 유전자는, N. 크라사가 미스칸터스 또는 아비셀에서 성장하였을 때 현저하게 증가된 발현을 나타낸 것으로 확인되었고, 이는 식물 세포벽 분해 산물(plant cell wall degradation products)이 세포 내로의 수송에 대한 이들의 관여를 암시한다. 생체 외에서의 셀룰라아제 및 헤미셀룰라아제에 의한 주요한 분해 산물(degradation products)은 셀로비오스, 글루코스, 자일로비오스(xylobiose) 및 자일로스이다. 이러한 운반체의 몇몇은 기능적으로 불필요할 수 있거나, 올리고당(oligosaccharides)을 수송할 수 있다. 이러한 추정상의 운반체(putative transporters)의 기능을 보다 더 분석하였다(실시예 7-9 참조). N. 크라사 운반체의 이형 발현(heterologous expression)에 의한 올리고당을 수송할 수 있는 다운스트림 과정(downstream processing strains)의 스트레인의 구성(Construction)은 바이오매스 가수분해 산물(biomass hydrolysis products)의 산업상의 발효(industrial fermentation)를 개선시킬 수 있다. 이러한 운반체의 중 어떠한 것 또는 그들이 수송할 수 있는 어떠한 것도 어느 곰팡이(any filamentous fungi)에서의 분자 또는 기능적인 수준(molecular or functional level)으로 특징지을 수 없다.
미스칸터스 및 아비셀에서 성장하는 동안 증가된 발현수준을 나타내는 많은 유전자는 그 밖의 섬유소분해성 균주(other cellulolytic fungi)에서 보존된 알려지지 않은 기능의 단백질을 코드한다. 16 가지의 스트레인의 표현형 만을 평가함으로써, 셀룰라아제 활성도에 현저한 영향을 주는 알려지지 않은 기능(unknown function)의 단백질을 코딩하는 유전자에서의 돌연변이체가 확인되었다. N. 크라 사에서 게놈의 자원(genomic resources)의 잘 알려진 유전학(well-understood genetics) 및 효용성(availability)은, 이러한 단백질의 생물학적 기능(biological function)뿐만 아니라 셀룰라아제 및 헤미셀룰라아제 생산의 조절적인 측면(regulatory aspects)을 결정하고, 식물 세포벽의 분해에 포함되는 세포외 효소 사이의 불필요한 중복 및 시너지(synergies)를 분석하기 위한 이상적인 유기체의 모델(ideal model organism)을 만들 수 있고,
미스칸터스에서
성장하는 동안의 상향조절된(
Upregulate
) 유전자의 돌연변이체의 스크리닝(
Screening
)
전사 프로파일링 실험(transcriptional profiling experiment)에서 확인된 추가적인 유전자를 분석하기 위해, 16 시간 동안 미스칸터스에서 성장한 네우로스포라에서 상향조절된 188 가지 유전자의 돌연변이체 표현형(phenotypes)을 분석하였다(실시예 1 참조). 각각의 유전자의 넉아웃 돌연변이체(knockout mutant)를 최소 Vogel's 배지에서 10-14 일 동안 성장시켰다. 분생자(Conidia)를 2 mL ddHiO 와 함께 채취하였고 mL 당 106 분생자의 농도로 250 mL 의 플라스크에 100 mL 배지 내에서 접종하였다(inoculated). 상이한 탄소원 중의 하나를 각각의 플라스크에 첨가하였다 : 2 % 수크로스, 2 % 아비셀 또는 2 % 미스칸터스(미국 캘리포니아 버클리의 캘리포니아 대학의 Calvin Laboratory 로부터 입수한 1mm 입자). 배양물(Cultures)을 4 일 동안 250 rpm 으로 교반시키면서 25 ℃ 에서 성장시켰다.
표 10 은 돌연변이체의 표현형을 리스트로 작성하였고, 이는 야생형과 비교하여 아비셀 또는 미스칸터스에서의 셀룰라아제 활성도 및 성장에서 중요한 차이점을 나타낸다. 아비셀 또는 미스칸터스에서의 성장은 "+" 점수체계(scoring system)로 평가하였다. 야생형의 성장은 "++" 로 나타내었다. 배양 상청액에서 전체 단백질을 Bradford 검정으로 측정하였고(900 μl Bradford dye 에 대한 100 μl 상청액), 엔도글루카나아제 활성도는 Megazyme 의 엔도글루카나아제 활성도의 퍼센트로서 표 10 에 나타내었다. 전체 셀룰라아제 활성도는 실시예 4 에 나타낸 바와 같은 상청액에서 셀로비오스 수준의 발견에 의해 측정하였다. 결과를 야생형의 퍼센트로서 표 10 에 나타내었다.
[표 10]
운반체 유전자의 추가적인 분석
실시예 1 에 기재된 바와 같이, 네우로스포라가 미스칸터스 및 아비셀에서 성장하였을 때 예측 당 운반체 단백질을 코딩하는 10 가지 유전자는 발현 수준이 증가됨을 나타내었다 : NCU00801, NCU00988, NCU01231, NCU04963, NCU05519, NCU05853, NCU05897, NCU06138, NCU08114 및 NCU10021. 이러한 유전자 중의 9 가지에 대한 결실 스트레인은 Fungal Genetics Stock Center 로부터 입수가능하다. NCU10021 의 결실 스트레인은 입수가능하지 않다.
NCU05853, NCU05897 또는 NCU08114 의 결실 돌연변이는 미스칸터스 또는 아비셀에서 성장 결함(growth defect)을 나타내는 스트레인 및/또는 셀룰라아제 효소 결함(cellulase enzyme defect)을 갖는 스트레인을 야기한다(실시예 6 ; 표 10 참조). △NCU05853 는 야생형과 비교하여 아비셀에서의 감소된 성장을 나타내었고 감소된 엔도글루카나아제 활성도를 나타내었고, △NCU08114 는 야생형과 비교하여 아비셀에서 감소된 성장을 나타내었고, 감소된 셀로비오스 수준을 나타내었다. 특히, 스포로트리쿰 서모필(Sporotrichum thermophile)의 발현 분석과 비교하였을 때, 다른 사상균(filamentous fungus), NCU05853(ST8454) 및 NCU08114(ST5194)의 상동성(homologs)은, S. 서모필은 글루코스와 비교하여 아비셀에서 성장하였을 때, 또한 상향조절되었고(upregulated)(실시예 8, 표 11 참조), 이는 추가적으로 셀룰로스 이용(cellulose utilization)에서 이들의 중요성을 나타낸다.
[표 11]
각각의 예측된 운반체의 기질의 동일성(identity)을 줄이기 위해서(narrow down), NCU05853 또는 NCU08114 의 결실 돌연변이를 포함하는 스트레인은 글루코스, 셀로비오스, 자일란 및 아비셀에서 배양하였다(표 12). 배양 배지는 2 % 의 탄소원에 Vogel's 배지를 포함한다. 둘 다의 돌연변이체는 아비셀에서 크게 감소되었지만 자일란, 글루코스, 자일로스 또는 셀로비오스에서는 그렇지 않았다.
[표 12]
헤미셀룰로스의 이용에서 이러한 운반체의 역할을 조사하기 위해, 네우로스포라가 자일란에서 성장하였을 때, 10 가지 운반체 유전자의 발현을 실험하였다. 스트레인을 2 % (w/v) 자일란을 갖는 Vogel's 염에서 성장시키는 것을 제외하고 실시예 4 에 기재된 바와 같은 방법을 사용하였다. 모든 10 가지 운반체의 발현은 자일란에서 성장하는 동안 상향조절되었고(upregulated)(표 13), 이는 그들인 헤미셀룰로스 분해로부터 유도된 당(예를 들어, 자일로비오스, 자일로스, 아라비노스, 자일로-올리고당) 뿐만 아니라 셀룰로스 분해로부터 유도된 당(예를 들어, 셀로비오스, 글루코스, 셀로-올리고당)을 운반할 수 있다. 돌연변이체 성장의 결과 및 발현 분석은 적어도 두 가지의 예측된 운반체 NCU05853 및 NCU08114 는 이당류(셀로비오스, 자일로비오스) 및/또는 올리고당(셀로덱스트린)을 운반할 수 있다.
[표 13]
다양한
탄소원에서
성장하는 동안의 N.
크라사
운반체의
스포로트리쿰
서
모필
상동성의
발현 분석
상이한 사상균(filamentous fungus)으로부터의 상동 유전자의 발현을 비교하기 위해, 스포로트리쿰 서모필의 발현 프로파일(expression profile)을, 글루코스, 아비셀 또는 코튼(cotton)에서 성장한 배양물(cultures)로부터 분석하였다. cDNA 는 글루코스, 아비셀 또는 코튼의 탄소원(carbon source)을 갖는 최소배지에서 16-30 시간 동안 성장시켰다.
첫째로, S. 서모필 게놈(genome)에서 네우로스포라 운반체 단백질의 상동성을 확인하기 위해서, 각각의 네우로스포라 서열은 BLAST 에서의 S. 서모필 단백질의 데이터베이스와 대비하여 비교하였다. 그리고 난 다음에 이러한 방법으로 발견된 S. 서모필 단백질의 서열을 BLAST 에서의 네우로스포라 단백질의 데이터베이스와 비교하였다. 이러한 결과를 도 10 에 나타내었다. 확인된 추정상의(putative) 네우로스포라 운반체의 모든 S. 서모필 상동성에 대해 아미노산 서열을 SEQ ID NOs: 22-32 에서 나타내었다.
다음으로, S. 서모필 상동성의 발현 프로파일을 실험하였다. 이러한 데이터를 표 11 에 나타내었다. 첫 번째 행(column)은 Joint Genome Institute S. 서모필 어셈블리(assembly)로부터 명명된 S. 서모필을 나타낸 것이다. 두 번째 행은 네우로스포라에서의 가장 밀접하게 관련된 추정상의 운반체에 대한 NCU 번호를 나타낸 것이다. 세 번째 행은 뉴클레오티드(nucleotides)에서 S. 서모필 유전자의 유전자 길이(gene length)를 나타낸 것이다. 네 번째 내지 여섯 번째 행은 탄소원(carbon source)으로서 2 % 의 글루코스, 아비셀 또는 코튼 볼(cotton balls)로 보충된 Vogel's 최소배지에서 성장하는 동안 발현수준[완전 발현 수준과 비교한, 리드(reads)의 번호]을 나타낸 것이다. 7 번째 내지 9 번째 행은 정규화된 발현 데이터를 나타낸 것이다[데이터 세트(dataset)에서 전체 리드(total reads)에 의해 나뉘어진 # 의 리드]. 마지막 두 개의 행은 아비셀/글루코스 또는 코튼/글루코스의 비율로서 각각의 유전자에 대한 상대적인 발현수준 데이터를 나타낸 것이다. NCU5853, NCU8114 및 NCU0801 의 상동(Homologs)은 아비셀 및 코튼 둘 다에서 성장하였을 때 상향조절되었다(upregulated). NCU6138 의 상동은 코튼에서 성장하였을 때 상향조절되었고, NCU4963 의 상동은 아비셀에서 성장하였을 때 상향조절되었다. 이러한 데이터는, 추정상의 운반체 NCU5853, NCU8114, NCU0801, NCU6138 및 NCU4963 은 셀룰로스의 이용(utilization)에서 중요한 역할을 함을 추가로 나타낸다.
셀로덱스트린
운반체의 분석 및 확인
순수한 셀룰로스에서 성장시켰을 때, N. 크라사는 세포내 β-글루코시다아제 뿐만 아니라 7 가지 주요한 촉진 상과 당 운반체(Major Facilitator Superfamily sugar transporters)의 전사를 증가시키는 것으로 나타냈다(Ex. 1; also see Supplemental Data, Dataset SI, page 6 in Tian 등의, PNAS, 2009). 특히, 이러한 세트(set)로부터의 각각의 운반체가 결실된 넉아웃 스트레인(knockout strains)은 결정형 셀룰로스에서 보다 서서히 성장하였고, 이는 이들이 셀룰로스계 조건(cellulolytic conditions) 하에서 셀로-올리고당의 흡수(cello-oligosaccharide uptake)에서 직접적인 역할을 함을 암시한다(Ex. 7; 표 10, 12). 예를 들어, NCU08114 의 결실은 매우 늦은 N. 크라사 성장 발달(도 11) 및 셀로비오스의 감소된 소비(도 12-13)를 야기하였다. 이러한 예에서, 운반체 유전자 NCU00801/cbt1 및 NCU08114/cbt2 는 셀로덱스트린의 운반체를 코드하기(encode) 위해 추가적으로 분석하고 확인하였다.
개별적으로 각각의 운반체의 기능을 검정하기(assay) 위해, 셀로비오스가 S. 세레비시아에 의해 대사 작용으로 분해되지(catabolize) 않고, 이들의 세포질(cytoplasm)에서 축적되지 않은 사실을 이용하였다(도 14). 세포내 β-글리코시다아제와 함께 기능적인 셀로비오스 운반체의 발현은, 셀로비오스가 단일 탄소원(sole carbon source)으로서 나타내었을 때 S. 세레비시아의 성장을 가능하게함을 판단할 수 있다. 효모 스트레인은, 녹색 형광 단백질(Green Fluorescent Protein, GFP)와 융합된 운반체 NCU00801 또는 NCU08114, 및 추정상의 세포내 β-글루코시다아제를 발현시키도록 유전자 조작되었다(engineered). 둘 다의 운반체는 발현되었고, 원형질막(plasma membrane)에 정확하게 위치하였다(도 15). NCU00801 또는 NCU08114 가 발현된 스트레인은 효모가 각각 0.0341 hr-1 및 0.0131 hr-1 의 특정한 성장률(growth rates)을 갖으면서 성장을 할 수 있게 한다(도 16A). 이러한 성장률은 글루코스에서의 각각의 30 % 및 12 % 의 성장률과 일치한다(도 17). 성장은 0.0026 hr- 1 의 비율로 성장한 추정상의 세포내의 β-글루코시다아제만이 발현되는 스트레인으로서, 셀로비오스에서 글루코스의 세포외 가수분해 후의 수송에 의해 설명되지 않고(도 16A), 대규모의 배양물(large-scale cultures)에서 성장하지 않았다(도 18). 이러한 관찰을 기반으로 CBTl 및 CBT2 로 명명된 NCU00801 및 NCU08114 은 셀로비오스 운반체로서의 기능에 대해 결정하였다.
운반체 기능을 직접적으로 검정하기 위해, 효모 세포 내로의 [3H]-셀로비오스의 흡수(uptake)를 측정하였다. CBTl 및 CBT2 둘 다는 각각 4.0 ± 0.3 μM 및 3.2 ± 0.2 μM 의 Km 값을 갖는 높은-친화도(affinity)의 셀로비오스 운반체를 발견하였다(도 19). CBT1 의 발현-정규화된(expression-normalized) Vmax 는 CBT2 의 2.2 배이고, 효모 성장 검정(yeast growth assays)에서 나타낸 차이점을 설명한 사실이다. 특히, 셀로비오스보다 더 길게 셀로덱스트린 분자는 cbt1 및 cbt2 가 발현된 효모의 성장을 지원하고(support)(도 20 ; 도 16B), 이는 셀로덱스트린 분자가 CBT1 및 CBT2 에 의한 수송됨을 나타낸다. 조화되어, CBTl 및 CBT2 에 의한 셀로비오스 수송은 과량의 셀로비오스에 의해 저해되고, CBT1 활성도는 셀로테트라오스(cellotetraose)에 의해 또한 저해된다(도 21). 게다가, 정제(purification) 상에서, β-글루코시다아제, NCU00130(도 22)는 셀로비오스, 셀로트리오스(cellotriose) 및 셀로테트라오스(cellotetraose)를 가수분해하는 것으로 발견하였다(도 16C).
cbt1 및 cbt2 의 오솔로그(Orthologs)는 균류의 계(fungal kingdom)에 폭넓게 분포됨을 확인하였고, 발견하였다(도 23). 최근의 발현 데이터는 균류(fungi) 및 식물 사이에 다양한 상호작용에서 이들이 중요한 역할을 함을 나타낸다. 예를 들어, 자낭기(ascomycete), 투버 멜라노스포룸 ( Tuber melanosporum ), 또는 담자균(basidiomycete), 락카리아 비컬라(Laccaria bicolor)는 엑토마이코리자(ectomycorrihzas)를 형성하기 위한 루트 팁(root tips)과 공생하여 상호작용하고(interacts), cbt1 의 오솔로그는 둘 다에서 상향조절되었다(Martin 등의., 2010). 마찬가지로, 사물기생군(saprophytes), 아스페르길루스 오리재(Aspergillus oryzae )(Noguchi 등의, 2009), 포스티아 플라센타(Vanden Wymelenberg 등의, 2010), 및 파네로캐츠 크라이소스포리움( Phanerochaete chrysosporium )(Vanden Wymelenberg 등의 , 2010)은, 식물 벽 물질과 접촉하였을 때 cbt2 의 오솔로그를 상향조절하였다. 클루이베로마이세스 락티스( Kluveromyces lactis) 및 피치아 스티피티스( Pichia stipitis )와 같은 특정 효모는 셀로비오스에서 성장하였고(Freer, 1991; Preez 등의, 1986), 셀로비오스 운반체는 클라비스포라 루시타니아( Clavispora lusitaniae )에서 보고되었다(Freer and Greene 1990). 이러한 연구에서, 이러한 효모의 모두는 cbt1, cbt2 의 오솔로그 또는 둘 다를 함유됨을 알아냈다(방법에 대해 하기 참조). 셀로비오스 운반체는 히포크레아 제코리나( Hypocrea jecorina )(트리코데르마 레세이 , Trichoderma reesei )에서 관찰하였지만, 운반체를 확인하지 않았기 때문에, 만약 이러한 활성도가 cbt1 또는 cbt2 의 오솔로그에 의할 수 있기 때문에 이는 명확하지 않다(Kubicek 등의., 1993).
셀룰로스계 균류(cellulolytic fungi)에 의한 셀로비오스 운반체의 용도(use)는, 그들이 셀룰로스에서 그들의 최적의 성장에 필수적임을 제시한다. 셀로비오스 이화작용(cellobiose catabolism)은 효모 에탄올 생산을 개선시키는지 여부를 테스트하기 위해, 상기에 구성된(constructed) 효모 스트레인은 발효 조건(fermentation conditions) 하에서 성장시켰다. 약간의 최적화에 대해, N. 크라사로부터 복사된(ported) 완전한 셀로비오스 이화작용 경로(complete cellobiose catabolism pathway)를 갖는 효모는, 86 % 의 이론값(Bai 등의, 2008)인 0.47 의 에탄올 수득률을 갖는 셀로비오스에서 에탄올로 유효하게 발효시킴을 나타내었다(도 24A). 이는 90-93 % 의 글루코스로부터 산업적인 수득률(industrial yields)과 비교할 만하다(Basso 등의, 2008). 셀로비오스에 대한 CBTl 및 CBT2 의 높은 친화도(high affinity)는 S. 세레비시아의 육탄당 운반체(hexose transporters)와 비교할 만하고(Reifenberger 등의, 1997), 세포외 β-글루코시다아제가 보고되어 있으며(Chauve 등의, 2010), 이는 셀로비오스/셀로덱스트린 수송 시스템(ellobiose/cellodextrin transport system)은 SSF 동안에 특히 유용함을 나타낸다. 예를 들어, 셀로비오스/셀로덱스트린 수송은 셀룰로스에서 글루코스로의 완전한 가수분해에 대한 요건을 보다 낮출 수 있고, 셀룰로스계 효소(cellulolytic enzymes)의 셀로비오스-매개 저해(cellobiose-mediated inhibition)를 감소시키며, 글루코스-의존적인 유기체(glucose-dependent organisms)에 의한 오염의 위험을 줄인다. 실제로, 셀로비오스/셀로덱스트린 수송 시스템이 발현되 효소는, 셀로비오스 및 글루코스 둘 다의 정상 상태의 농도(steady state concentration)를 감소시키고 에탄올 생산율을 증가시켜 SSF 반응의 효율을 현저하게 개선시킨다(도 24B, C).
셀룰로스로부터의 바이오연료(Biofuel)의 생산은 개선된 숙주 스트레인(host strains)에 의한 연료 생산과 함께 식물 바이오매스(plant biomass)에서 당으로의 효율적이고 경제적인 디폴리머라이제이션(depolymerization)을 필요로 한다(Kumar 등의, 2008). 셀룰로스계 균류는 식물 바이오매스에서의 최적의 성장에 대한 셀로-올리고당 수송 경로를 사용하는 것을 본원에서 보여주고 있다. 게다가, 효모에서 이러한 경로의 재구성(reconstitution)은, 그들이 운반체 및 세포내의 셀로-올리고당 가수분해 효소(cello-oligosaccharide hydrolase)로 구성된 최소한의 경로를 갖는, 셀로비오스 이화작용(cellobiose catabolism)을 증진시키기 위한 모듈식의 방식(modular fashion)으로 복사될 수 있다(ported)(도 25). 효모 및 다른 유기체의 바이오연로-생산 스트레인(biofuel-producing strains)에서 셀로덱스트린 수송의 용도(use)는 보다 경제적이고 실행가능한 셀룰로스계 바이오연료 공정(cellulosic biofuel processes)을 생산하는데 대단히 중요하다.
운반체 및 β-글리코시다아제
오솔로그
셀룰로덱스트린 운반체에 대한 GenBank 접근번호(accession numbers) 또는 Joint Genome Institute (JGI) 단백질 ID (PID) 번호는 하기와 같다 : 투버 멜라노스포룸( Tuber melanosporum ) , CAZ81962.1 ; 피치아 스티피티스 ( Pichia stipitis ), ABN65648.2 ; 락카리아 비컬라( Laccaria bicolor ) , EDR07962 ; 아스페르길루스 오리재(Aspergillus oryzae ), BAE58341.1 ; 파네로카에테 크라이소스포리 움( Phanerochaete chrysosporium ), PID 136620(JGI)(Martinez 등의, 2004); 포스티아 플라센타(Postia placenta ), PID 115604(JGI)(Martinez 등의, 2009). 사카로미 세스 세레비시아 ( Saccharomyces cerevisiae ) HXT1 및 클루이베로마이세스 락티 스( Kluyveromyces lactis ) LACP 에 대한 GenBank 접근번호는 각각 DAA06789.1 및 CAA30053.1 이다. P. 크라이소스포리움 및 P. 플라센타 게놈은 각각 genome.jgi-psf.org/Phchrl/Phchrl.home.html 및 genome.jgi-psf.org/Pospll/Pospll.home.html 에서 접속할 수 있다.
NCU00130 의 오솔로그인 셀로덱스트린 가수분해 효소에 대한 GenBank 접근번호는 하기와 같다 : T. 멜라노스포리움 ( T. melanosporum ), CAZ82985.1 ; A. 오리재(A. oryzae), BAE57671.1; P. 플라센타 (P. placenta ), EED81359.1 ; 및 P. 크라이소스포리움 (P. chrysosporium), BAE87009.1. 셀로덱스트린 운반체 오솔로그를 포함하는 그 밖의 유기체는 GH3 패밀리에서의 유전자를 세포내 β-글리코시다아제인 것으로 예측된 GH3 패밀리에서의 유전자를 포함하며(Bendtsen 등의, 2004 ; Cantarel 등의, 2009), 하기와 같다 : 클루이베로마이세스 락티스 ( Kluyveromyces lactis ), CAG99696.1 ; 락카리아 비컬라( Laccaria bicolor ), EDR09330 ; 클라비스포라 루시타니아 ( Clavispora lusitaniae ) , EEQ37997.1 ; 및 피치아 스티피티스 ( Pichia stipitis ), ABN67130.1.
스트레인 및 배지(
Media
)
본 연구에서 사용된 효모 스트레인은 하기와 같은 유전자형(genotype)을 갖는 YPH499 이다(Sikorski 등의, 1989) : MATa ura3 -52 lys2 -801_ amber ade2 -101_ochre trpl -△63 his3 -△200 leu2 -△l. 100 mg/L 아데닌 헤미설페이트(adenine hemisulfate)로 보충된 YPD 배지에서 성장시켰다. 변형된 스트레인(Becker 등의, 2001)은, 100 mg/L 아데닌 헤미설페이트(adenine hemisulfate)로 보충된 적절한 완전한 최소 드롭아웃 배지(complete minimal dropout media)에서 성장시켰다. 본 연구에서 사용된 네우로스포라 크라사 스트레인은 Fungal Genetics Stock Center 로부터 입수하였고(McCluskey 2004), WT(FGSC 2489) 및 두 가지의 셀로비오스 운반체 결실 스트레인[FGSC 16575, ANCU00801.2 및 FGSC 17868, ANCU08114.2 (Colot 등의, 2006)]을 포함한다.
플라스미드 및
클로닝
프라이머(primers)-ATATATGAGCTCGTGAGTAAGGAAAGAGTGAGGAACTATC(SEQ ID NO : 53) 및 ATATATACTAGTTGTTTTATATTTGTTGTAAAAAGTAGATAATTACTTCC(SEQ ID NO : 54)-를 사용한 SacI 및 SpeI 사이에 삽입된 S. 세레비시아 PGK1 프로모터를 포함된 변형을 갖는 2 μ의 플라스미드, pRS426 내로 운반체를 클론하였다(cloned). [상기와 하기의 모든 프라이머에서, 제한 부위(restriction sites)는 밑줄을 그었다]. C-말단 Myc-tag 및 최적화된 Kozak 서열을 갖는 NCU00801 (Miyasaka 1999)을, 프라이머-ATGGATCCAAAAATGTCGTCTCACGGCTCC(SEQ ID NO: 55) 및 ATGAATTCCTACAAATCTTCTTCAGAA ATCAATTTTTGTTCAGCAACGATAGCTTCGGAC(SEQ ID NO: 56) -를 사용하여 BamHI 및 EcoRI 사이에 삽입하였으며, C-말단 Myc-tag 및 최적화된 Kozak 서열을 갖는 NCU08114 을, 프라이머-ATACTAGTAAAAATGGGCATCTTCAACAAGAAGC(SEQ ID NO : 57) 및 GCATATCGAT CTACAAATCTTCTTCAGAAATCAATTTTTGTTCAGCAACAGACTTGCCCTCA TG(SEQ ID NO: 58)-를 사용하여 SpeI 및 ClaI 사이에 삽입하였다. GFP 융합(fusions)을 생산하기 위해, Gly-Ser-Gly-Ser 의 N-말단 링커(linker)를 갖는 슈퍼폴더 GFP(superfolder GFP)(Pedelacq 등의, 2006)를, 프라이머-TATTAAATCGATGGTAGTGGTAGTGTGAGCAAGGGC GAGGAG(SEQ ID NO: 59) 및 TATTAAGTCGACCTACTTGTACAGCTCGTCCATGCC(SEQ ID NO: 60)-를 갖는 PGK1 프로모터- 함유하는 pRS426 플라스미드(PGK1 promoter- containing pRS426 plasmid)를 ClaI and SaiI 사이에 첫 번째로 삽입하였다. 운반체는 GFP 와 융합된 다음에 하기와 같다 : 프라이머-NCU00801 은 CATGGATCC ATGTCGTCTCACGGCTCC(SEQ ID NO: 61) 및 TATAATGAATTCAGCAACGATAGCTTCGG AC(SEQ ID NO: 62)-를 사용하여 BamHI 및 EcoRI 사이에 삽입되었으며, NCU08114 은 TATTAAACTAGTATGGGCATCTTCAACAAGAAGC(SEQ ID NO: 63) 및 TTATAAGAATTCAGCAAC AGACTTGCCCTCATG(SEQ ID NO: 64) 프라이머를 사용한 SpeI 및 EcoRI 사이에 삽입되었다.
β-글리코시다아제, NCU00130 을 상기한 바와 같이 PGK1 프로모터를 포함한 변형된, 2 μ의 플라스미드, pRS425 내로 클론시켰다. 최적화된 Kozak 서열 및 C-말단 6xHis tag 를 갖는 NCU00130 을, 프라이머-GCATACTAGTAAAAATGTCTCTT CCTAAGGATTTCCTCT (SEQ ID NO: 65) 및 ATACTGCAGTTAATGATGATGATGATGATGGTCCTTC TTGATCAAAGAGTCAAAG (SEQ ID NO: 66)-을 사용한 SpeI 및 PstI 사이에 삽입시켰다. 모든 구조물은 Xhol 및 Kpnl 사이의 Cyc 전사 종결자(transcriptional terminator)를 포함한다. 모든 N. 크라사 유전자는, 단일 탄소원(sole carbon source)으로서 순수한 셀룰로스(아비셀)을 갖는 최소배지(minimal media)에서 배양된 N. 크라사(FGSC 2489)로부터 분리된 mRNA 로부터 합성된 cDNA 로부터의 PCR 에 의해 증폭되었다.
효모 성장 검정
셀로-올리고당(cello-oligosaccharides)에서의 성장을 모니터하기 위해, 유전자조작된 스트레인을 적절한 드롭아웃(dropout)을 갖는 5 mL 의 완전한 최소배지에서 밤새 성장시켰다. 이러한 스타터 배양물(starter cultures)을 25 mL 의 ddH20 로 3 번 세척하였고, 적절한 완전 보충 배지(Complete Supplemental Media, CSM) 및 1 % (w/v)의 셀로비오스 또는 0.5 % (w/v)의 셀로트리오스 또는 셀로테트라오스를 더한 효모 질소 염기(Yeast Nitrogen Base, YNB)에 0.1 의 OD(600 nm 에서)에 재현탁시켰다(resuspended). 30 ℃ 에서 최대한의 진폭으로 일정하게 교반시키고 0.4 mL 의 최종 검정 부피를 갖는 Bioscreen CTM 에서 검정을 실시하였다. OD 에서의 변화는 600 nm 에서 또는 450 - 580 nm 의 광대역필터(wideband filter)를 사용하여 측정하였다. 성장률을 각각의 성장 곡선의 선형 부분(linear portion)으로부터 획득하였고, 세 가지 독립적인 실험의 평균 ± 실험들 사이의 표준편차로서 나타내었다. 셀로트리오스 및 셀로테트라오스를 Seikagaku Biobusiness Corporation (일본 도교)로부터 입수하였다.
NCU00130 의
정제 및 이의 활성도의 검정
cbt1 및 NCU00130 이 발현된 S. 세레비시아의 1L 배양물을 완전 최소 배지에서 2.0 의 OD 에서 성장시켰다. 세포를 원심분리하여 채취하였고, 30 mL 의 세포용혈 완충용액(lysis buffer)[50 mM NaH2P04(pH 8.0), 300 mM NaCl, 10 mM 이미다졸, 2 mM p-ME, CompleteTM Mini, EDTA 유리된 분해효소 저해제 혼합물(free protease inhibitor cocktail)]에 재현탁시켰다. 세포를 음파처리(sonication)하여 용해하였고, 용해물(lysate)을 30 분 동안 15,000 g 에서 원심분리하여 투명하게 하였다(cleared). 용해물을 중력 유동(gravity flow)으로 1 mL 의 니켈-NTA 수지(nickel-NTA resin)에 결합시켰고, 25 mL 세척 완충용액(wash buffer)(세포용혈 완충용액과 동일하지만, 20 mM 의 이미다졸을 갖음)으로 세 번 세척하였다. NCU00130 은 5 mL 의 용리 완충용액(elution buffer)으로 용출시켰고(eluted), 적절한 분획(fractions)을 모으고(pooled), 저장 완충용액(storage buffer)[인산 완충 식염수(PBS), 2 mM DTT, 10 % 글리세롤] 내로 교환시키고(exchanged), 앨리쿼트하고(aliquoted), 액체 질소에 냉동시키고, -80 ℃ 로 저장하였다. 순도(Purity)는 SDS-PAGE 에 의해 측정되고(도 22), 단백질 농도는 108,750 M-1cm- 1 의 흡광계수(extinction coefficient)를 사용하여 280 nM 에서의 흡광도로부터 측정하였다.
정제된 NCU00130 을, 상이한 셀로덱스트린 기질을 갖는 가수분해 활성도(hydrolysis activity)로부터 검정하였다. 3 mM DTT 가 더해진 150 μL PBS 에서 500 μM 의 각각의 당과 함께 5 pmol 효소를 배양하여 측정하였다. 100 μL 를 제거하기 전에 30 ℃ 에서 40 분 동안 반응을 진행시키고, 400 μL 의 0.1 M NaOH 로 퀀칭하였다(quenched). CarboPac PA200 컬럼으로, Dionex ICS-3000 를 갖춘 이온 크로마토그래피로 결과를 분석하였다. 피크(Peak)를 전기화학 검출기(electrochemical detector)로 발견하였다.
운반체
오솔로그(
Orthologs)
의
계통 분석(
Phylogenetic
Analysis
)
CBTl 및 CBT2 의 오솔로그의 아미노산 서열을 온라인 데이터베이스(online databases)로부터 수득하였다. 다수의 서열 정렬(Multiple sequence alignments)을 T-Coffee 를 사용하여 수행하였다(Notredame 등의, 2000). 최대한의 가능성을 갖는 계통발생론(maximum likelihood phylogeny)을 100 부트트랩스(Bootstraps)을 갖는 PhyML 버전 3.0(Guindon and Gascuel 2003)을 사용하여 측정하였다. 둘 다의 프로그램을 Phylogeny.fr(웹페이지 phylogeny.fr/)을 통해 접속하였다. 결과적으로 생성된 트리(tree)는 FigTree v. 1.2.1 으로 나타내었다(웹페이지 tree.bio.ed.ac.uk/).
발효(
fermentation
) 및
SSF
발효 및 SSF 실험에서, NCU00130 가 발현된 효모, 및 Myc-태그된(tagged) cbt1 또는 운반체가 없는 것 사이에 비교를 실시하였다. 이러한 스트레인은 완전 최소 배지에서 밤새 산소를 필요로하면서 성장하였고, 25 mL 의 물로 세 번 세척하였으며, 밀봉된 혈청 플라스크(sealed serum flasks)에서 적절한 CSM, 및 2% (w/v) 셀로비오스 또는 3% (w/v) 순수한 셀룰로스(아비셀) 둘 중의 하나를 더한 50 mL 의 YNB 에서 2.0 의 최종 OD 에 재현탁시켰다. SSF 반응물은 β-글루코시다아제 보충(supplementation) 없이 필터-멸균된(filter-sterilized) Celluclast(Sigma C2730)의 50 필터페이퍼 유닛(Filter Paper Units)/g 셀룰로스를 또한 포함한다. 반응물은 교반시키면서 30 ℃ 에서 산소결핍에 의해(anaerobically) 실행하였다. 나타낸 시점에서, 1 mL 의 샘플을 제거하였고, 0.2 ㎛ 의 시린지 필터(syringe filter)를 통해 여과하였다. 여과액(filtrate)에서의 에탄올, 글루코스 및 셀로비오스 농도는 Aminex HPX-87H 컬럼 및 굴절률 검출기(refractive index detection)을 갖는 HPLC 에 의해 측정하였다.
N.
크라사
성장 및
알라마
블루
®
검정(
Alamar
Blue
Assays
)
WT N.크라사(FGSC 2489) 및 동종핵성(homokaryotic) NCU08114(FGSC 17868) (Colot 등의, 2006)는 Fungal Genetics Research Center(McCluskey 2003)로부터 입수가능하고, 250 mL 의 언배플드 플라스크(unbaffled flask)에 2 % 의 수크로스 또는 순수한 셀룰로스(아비셀)을 더한 50 mL 의 Vogel's 염에서 25 ℃ 에서 성장시켰다. 16 시간 또는 28 시간 후에, 각각의 100 μL 의 알라마블루®를 첨가하고, 배양물을 20 분 동안 실온에서 배양하였다. 이러한 시간에서 1 mL 의 샘플을 제거하고, 잔해(debris)를 펠렛화시키고(pelleted), 100 μL 의 상청액의 형광(fluorescence)을, Beckman Coulter Paradigm plate reader 에서 535/595 nm 의 여기/방출(excitation/emission) 파장으로 측정하였다.
N.
크라사
셀로비오스
수송 검정
WT N. 크라사(FGSC 2489), 및 NCU00801(FGSC 16575) 및 NCU08114(FGSC 17868)의 동종핵성 결실 라인(homokaryotic deletion lines)을 Fungal Genetics Stock Center 로부터 획득하였고(McCluskey 2003), 106 분생자(conidia)/mL 의 접종물(inoculum)을 시작으로, 25 ℃ 에서 2 % (w/v) 수크로스를 더한 50 mL 의 Vogel's 염에서 16 시간 동안 성장시켰다. 균사체(Mycelia)는 원심분리에 의해 재취하였고, Vogel's 염으로 세 번 세척하였고, 운반체 발현을 유도하기 위해 4 시간 동안 0.5% (w/v) 순수한 셀룰로스(아비셀)을 더한 Vogel's 염에 이동시켰다(transferred). 10 mL 의 배양물을 원심분리로 채취하고, Vogel's 염으로 세 번 세척하고, 시클로헥사미드(100 ㎍/mL) 및 90 μM 의 각각의 셀로덱스트린(셀로비오스, 셀로트리오스 또는 셀로테트라오스)을 더한 1 mL 의 ddH2O 에서 재현탁시켰다. 셀로덱스트린 소비를 측정하기 위해, 100 μL 를 15 분 후에 제거하고, 원심분리로 투명하게 만들고(clarified), 900 μL 의 0.1 M NaOH 내로 이동시켰다. 상청액에 남아있는 당의 양은 CarboPac PA200 컬럼을 사용한 Dionex ICS-3000 을 갖는 HPLC 로 측정하였다. 피크를 전기화학 검출기(electrochemical detector)로 감지하였다(detected).
GFP
형광 및
공초점
형광 현미경(
Confocal
Fluorescence
Microscopy
)
벌크-셀 GFP 형광(Bulk-cell GFP fluorescence)을 485/535 nm 의 여기/방출 파장으로 Beckman Coulter Paradigm plate reader 에서 측정하였다. 공초점 형광 현미경은, Metamorph 소프트웨어로 조절되고 488 nm 레이저를 갖는 Yokogawa CSU-X1 스피닝 디스크 헤드(spinning disc head)에 부착된 Leica SD6000 현미경에서 lOOx 1.4 NA 유침 대물렌즈(oil immersion objective)를 사용한 0.8 - 1.2 의 OD (600 nm 에서)에서의 세포를 가지고 실시하였다. Z 시리즈는 200 nm 스텝 사이즈(step size)로 기록하였고 Image J 를 사용하여 분석하였다.
[
3
H]
셀로비오스
수송 검정(
Transport
assays
) 및 동역학적 파라미터(
Kinetic
Parameters
)
수송 검정을 오일-스톱 방법(oil-stop method)의 변형(modification)을 사용하여 실시하였다(Arendt 등의, 2007). GFP 에 융합된 cbt1 또는 cbt2 가 발현된 효모 스트레인은 OD(선택 배지에서 1.5-3.0 의 600 nm 에서)에 대해 성장되었고, 차가운 검정 완충용액[30 mM MES-NaOH(pH 5.6) 및 50 mM 에탄올]으로 세 번 세척하였으며, 20 의 OD 에 재현탁시켰다. 수송 반응을 시작하기 위해, 50 μL 의 세포를, 100 μL 의 실리콘유(silicone oil)(Sigma 85419) 상에서 층으로 놓여진(layered) 50 μL 의 [3H] 셀로비오스에 첨가하였다. 반응물(Reactions)을 17,000 에서 1 분 동안 오일을 통해 세포를 회전시켜 중단시키고, 튜브를 에탄올/건조 아이스에서 냉동시키고, 세포-펠렛(cell-pellets)을 함유하는 튜브-바닥(tube-bottoms)을 1 mL 의 0.5 M NaOH 내로 잘라내었다(clipped off). 펠렛(pellets)을 밤새 용해시키고, 5 mL 의 Ultima Gold 신틸레이션 유체(scintillation fluid)를 첨가하고, CPM 을 Tri-Carb 2900TR 신틸레이션 계수기에서 측정하였다. [3H] 셀로비오스는 Moravek Biochemicals, Inc.로부터 구매하였고, 4 Ci/mmol 의 고유활성도(specific activity) 및 순도 > 99 % 을 갖는다. 동역학적 파라미터는 셀로비오스 농도의 범위에 대한 3 분 이상 [3H] 셀로비오스 흡수의 선형 비율(linear rate)로 측정하여 결정하였다. Vmax 및 Km 값은 SigmaPlot® 에 의한 비선형회귀(non-linear regression)에 의한 셀로비오스 농도 대 비율의 구성(plot)에 대한 단일 사각형, 2-파라미터의 쌍곡선 함수(single rectangular, 2-parameter hyperbolic function)에 적용시켜 결정하였다. Vmax 값은 수송 검정을 시작하기 전에 즉시 OD 200 에서 100 μL 의 세포로부터의 GFP 형광(fluorescence)을 측정하여 운반체의 존재비(abundance)에서의 차이점에 대해 정규화시킨 것이다. 본문에서 나타낸 동역학적 파라미터는 세 가지 분리 실험으로부터의 평균 ± 표준편차이다. 비교 검정은 250 μM 의 각각의 경쟁자(competitors)의 존재 하에서 20 초 이상 50 μM [3H]-셀로비오스의 수송을 측정하기 위해 실시하였다.
대규모 효모 성장(
Large
Scale
Yeast
Growth
)
상이한 탄소원에서의 성장을 모니터하기 위해, 유전자 조작된 스트레인을 밤새 적절한 드롭아웃(dropouts)으로 5 mL 의 완전 최소 배지(complete minimal media)에서 성장시켰다. 이러한 스타터 배양물(starter cultures)을 25 mL의 ddH20 로 세 번 세척하고, 적절한 완전 보충 배지(Complete Supplemental Media, CSM) 및 2 % (w/v) 셀로비오스를 더하여 50 mL 효모 질소 염기(Yeast Nitrogen Base, YNB)에 0.1 의 OD(600 nm 에서)에 재현탁시켰다(resuspended). 배양물을 200 rpm 으로 교반시키면서 30 ℃ 에서 250 mL 의 저해시키지 않는 플라스크에서 성장시켰다. OD(600 nm 에서)에서의 변화를 정기적으로 샘플을 제거하면서 모니터하였다.
셀로덱스트린
운반체 기능에 대한 중요한
잔기(Critical Residues)의
확인
본 실시예에서, 서열 분석 및 돌연변이유발(mutagenesis) 연구를 셀로덱스트린 운반체에서 보존되고(conserved) 기능적으로 중요한 잔기를 확인하기 위해 사용하였다. 게다가, 추가적인 셀로덱스트린 운반체를 확인하였다.
셀로덱스트린 운반체 NCU00801(cbt1) 또는 NCU08114(cbt2) 및 야생형 β-글루코시다아제 NCU00130 의 다양한 돌연변이체(mutants)가 발현된 효모 스트레인의 성장률을 탄소원으로서 셀로비오스와 함께 성장하였다. NCU00801 의 96 위치 및 NCU08114 의 96 위치에서의 아미노산 잔기는 생산자의 지시(manufacturer's instructions)에 따라 QuickChange® II 사이트-직접적인 돌연변이유발 키트(Site-directed Mutagenesis Kit)(미국 캘리포니아 라졸라 Stratagene 에서 입수)를 사용하여 알라닌(alanine)에 대해서 개별적으로 돌연변이시켰다(mutated). 스트레인을 2 % 셀로비오스를 갖는 합성 정의된 배지(synthetic defined media)-ura -leu 100 mg/L 아데닌에서 성장하였다. 배양물을 두 가지의 독립된 군집(colony)로부터 시작하였다.
도 26(A, B)에서 나타낸 결과가 나타낸 바와 같이, W66, L73, Y74, N87, Y89, D90, Q 104, F107, G113, F120, Y123, D139, G142, K144, M147, G150, Q169, F170, G173, R174, G178, G180, P189, Y191, E194, P198, R201, Y208, W235, R236, Q242, 257PESPRF262 (SEQ ID NO: 67), Y279, G283, E296, D307, K308, W310, D312, R325, G336, Y345, N369, D385, F462, P468, E476, T480, 또는 G486 에서의 치환(substitutions)을 갖는 NCU00801 이 발현된 돌연변이 스트레인은 야생형 스트레인과 비교하여 적어도 25 % 의 성장 결함(growth defect)을 나타내었다.
NCU08114 에서 알라닌 스캐닝 실험(alanine scanning experiment)은 기능적으로 중요한 바와 같은 하기의 잔기를 나타낸다 : L38, Y39, G54, D56, F73, G91, P100, D104, G107, R108, Ml 18, R139, F144, Q150, P154, E159, P163, H165, R166, Y173, N174, W199, Q214, 222PESP225 (SEQ ID NO: 68), Y244, H245, D249, E258, E268, Q302, W303, S304, N306, Y312, F359, L360, F402, Y403, S404, Y414, E417, P420, Y421, K426, N442, N446, P447, W459, K460, E482, T483, L488, E489, E490, D496, and G497 (도 26b).
특히, 모티프(motifs) 73LYF75, 257PESP260 (SEQ ID NO: 69) 및 278KYH280 [NCU00801 의 잔기 번호(residue numbering)]는 둘 다의 운반체[NCU00801의 잔기 257PESP260(SEQ ID NO: 69) 및 NCU08114 의 잔기 222PESP225(SEQ ID NO: 68)]에서 기능적으로 중요하게 보이는 것 같고, 이는 29 % 의 아미노산 서열 동일성을 갖는다(도 26B, C). 보통 운반체 또는 특히 (이중-밑줄쳐진) β-링크된 운반체에서 보존된 몇몇의 잔기(도 26B, C 에 이텔릭체)는 예를 들어 D90(NCU00801), D56(NCU08114), 및 L73(NCU00801), L38(NCU08114), 운반체 기능(밑줄쳐진)에 대해 중요함을 실험적으로 나타낸 것이다. 돌연변이 유발 실험(mutagenesis experiment)의 결과는 기능적으로 중요한, 예를 들어 Q168(NCU00801) 및 Q214(NCU08114)와 같은 NCU00801/NCU08114 계통군(clade)[캡드(capped)]에서 보존된 잔기를 또한 보여준다. 게다가, 본 실험에서 기능적으로 중요한 것으로 알려진 다수의 잔기는 S. 세레비시아 당 운반체(Hxtl/Hxt3), 예를 들어 L73(NCU00801) 및 L38(NCU08114)에서 보존되는 것으로 사전에 나타내었다.
상이한 유기체로부터의 N. 크라사 셀로덱스트린 운반체의 오솔로그(Orthologs)는 또한 연구되어있다(도 27). 대표적인 오솔로그는 Genescript 에 의해 합성하였고, 발현 벡터, 사이트(sites) BamHl 및 HindⅢ 를 사용한 Cupl 프로모터를 포함하는 pRS426 내로 클론되었다(cloned). 이러한 구성물은 세포내 β-글루코시다아제, NCU00130 와 함께 효모 스트레인 YPH499 내로 형질전환되었다(transformed). 셀로비오스가 단일 탄소원으로서 존재할 때, 이러한 스트레인의 성장률을 측정하여 운반체 활성도를 측정하였다.
그 대신에, 추정상의 오솔로그를 함유하는 상이한 진균(fungal) 스트레인은 셀로비오스로 보충된 리치 배지(rich media)에서 재배하였다. 전체 RNA 는 분리되었고, cDNA 내로 역전사되었다(reverse transcribed). 중합효소 연쇄 반응(PCR)은 cDNA 로부터 직접적으로 추정상의 운반체 유전자를 증폭시키는데 사용하였다. 그러나, 조절 메카니즘(regulation mechanism) 및 발현 패턴(expression pattern)은 균종(fungal species)에서 셀로덱스트린 운반체에 대해 잘 알려지지 않았기 때문에, 배양 조건(cultivation condition)의 변경에도 불구하고 추정상의 운반체를 코딩하는 cDNAs 를 항상 수득할 수 있는 것은 아니다. 이러한 경우에, 프라이머를 GenBank 로부터의 이에 상응하는 cDNA 서열에 따라 설계하였고(designed), 주형(template)으로서 게놈의(genomic) DNA 를 사용하여 엑손(exons)을 증폭시키는데 사용된다. 오버랩-연장 PCR(Overlap-extension PCR)은 전체 길이의 유전자(full-length genes) 내로 엑손을 모으는데 사용된다. DNA 조립 방법(DNA assembler method)을 사용한 HXT7 프라이머 및 HXT7 종결자(terminator)를 포함하는 pRS424 셔틀벡터(shuttle vector) 내로 결과적으로 생성된 PCR 산물을 클론하였다. 형질전환체(transformant)로부터 분리된 효모 플라즈마는 대장균 DH5a 내로 재형질전환되었고(retransformed), 분리된 대장균 플라즈마는, 원본(original) 운반체 유전자를 증폭시키는데 사용되는 프라이머를 사용한 진단 PCR(diagnostic PCR)에 의해 첫 번째로 점검되었다(checked). 전체적인 오픈리딩프레임(open reading frames)을 플라즈마의 정확한 구성을 확인하기 위한 배열 순서를 밝히기 위해(sequencing) 제출되었다. P. 스티피티스로부터의 오솔로그 LAC2, HXT2.1 및 HXT2.6 에서, 하나 또는 그 이상의 선택가능한 코돈(alternative codons)(CUG)은 Leu 를 Ser 로 치환하였다. 효모 상동의 재조합 매개 DNA 조립 방법(yeast homologous recombination mediated DNA assembler method)을 사용하여 대부분의 클로닝 작업(cloning work)을 수행하였다. pRS424-HXT7-GFP 플라스미드를 추정상의 셀로덱스트린 운반체의 클로닝(cloning)에 사용하였다. 이러한 플라스미드에서, HXT7 프로모터, 둘 다의 말단(both ends)에서 EcoRI 사이트를 갖는 측면에 배치된(flanked) GFP 유전자 및 HXT7 종결자(terminator)는 Clal 및 BamHI 에 의해 선 모양으로 만들어진 pRS424 셔틀 벡터(New England Biolabs) 내로 조립되었다. HXT7 프로모터 및 종결자(terminator)와 서열 동일성(sequence identity)을 공유하는(sharing) DNA 단편과 함께 측면에 배치된(flanked) 추정상의 운반체의 PCR 산물은 표준 리튬 아세테이트 방법(standard lithium acetate method)을 사용한 pRS424-HXT7-GFP 소화된(digest) EcoRI 를 갖는 CEN.PK2-1C 내로 공동으로 이동되었다(co-transferred). 결과적으로 생성된 형질전환 혼합물(transformation mixture)을 형질전환체(transformants)를 회복하기 위해(recover) 2 % D-글루코스로 보충된 SC-Trp 플레이트에 도말하였다(plated). 추정상의 셀로덱스트린 운반체 오솔로그가 발현된 효모 및 NCU00130 를 단일 탄소원으로서 셀로비오스에서의 성장을 테스트하였다.
추정상의 셀로덱스트린 운반체 오솔로그의 목록 및 본 연구로부터 수득한 결과를 표 14 에 나타내었다.
[표 14]
특정한 경우에, 클론된 오솔로그의 서열은 정확하게 결정되었고, 이러한 클론이 발현된 효모는 셀로비오스를 활용할 수 있었다. 따라서, 이러한 클론, 피치아 스티피티스로부터의 LAC2 및 아스페르길루스 클라바투스로부터의 XP_001268541.1 을 기능적인 셀로비오스 운반체임을 확인해주었다. 그 밖의 클론의 셀로비오스 수송 기능의 테스트는 여전히 진행 중이다(in progress). 데이터베이스에서 발행된 서열(the published sequences)과 상이한 서열을 갖는 클론된 오솔로그(Cloned orthologs)(예를 들어, 삽입, 결실 등을 갖는 하나)(표 14)는, 재-클론되고(re-cloned), 다시 배열 순서를 밝히고(re-sequences), S. 세레비시아에서 이들이 발현되고 성장률을 모니터하여 셀로비오스 운반체 활성도에 대한 유사하게 테스트되었다.
NCU00801, NUC08114 의 정렬(alignment) 및 이러한 운반체의 기능적인 오솔로그를 도 28 에 나타내었다. 도 28A 에서의 정렬은 추정적이고 확인된 셀로덱스트린 운반체를 둘 다를 포함하는 반면에, 도 28B 에서의 정렬은 오직 확인된 셀로덱스트린 운반체만을 포함한다. 게다가, 도 28C 는 NCU00801 및 NCU08114 의 정렬을 나타낸다. 두 개의 운반체는 29 % 의 아미노산 서열 동일성(identity)을 공유한다(share).
셀로덱스트린 운반체 기능에 대한 중요한 모티프(Motifs)는 당과 운반체 사이의 다수의 서열 정렬(multiple sequence alignments)의 육안검사(visual inspection)에 의해 확인되었다. 명확하게, 셀로덱스트린 운반체에 공통되는 모티프는 추정상의 셀로덱스트린 운반체 오솔로그 및 확인된 셀로덱스트린 운반체 사이의 T-COFFEE 에서 생성된 다수의 서열 정렬로부터 확인되었다. 이러한 모티프가 셀로덱스트린 운반체에 대해 대체로 특유한지를 확인하기 위해, S. 세레비시아의 육탄당 운반체, 인간 글루코스 운반체, Glut1 및 T-COFFEE 에서 생성된 두 가지의 N. 크라사 단당류 운반체 사이의 다수의 서열 정렬로부터 이들의 부재를 확인하였다.
확인된 모티프는 하기에 기재되었다. 모티프에서, NCU00801 의 기능에 중요한 것으로 발견된 잔기를 밑줄을 그었다. NCU08114 의 기능에 중요한 잔기를 상첨자(superscript) 로 표시하였다. 둘 다의 운반체의 기능에 중요한 잔기를 상첨자 "*" 로 표시하였다. 모든 모티프를 PROSITE 표기법(notation)을 사용하여 나타내었다. PROSITE 모티프를 읽는 방법의 예로서, 하기의 모티프 [AC]-x-V-x(4)-{ED}는 [Ala 또는 Cys]-어떤 것(any)-Val-어떤 것-어떤 것-어떤 것-어떤 것-{Glu 또는 Asp 외의 어떠한 것}({any but Glu or Asp}) (SEQ ID NO: 13)을 의미한다.
모든 당 운반체와 같은 셀로덱스트린 운반체는 12 개의 트랜스멤브레인 α-헬릭스(transmembrane α-helices)를 갖는다. 셀로덱스트린 운반체의 N- 및 C-말든은 모두 세포내(both intracellular)이다.
트랜스멤브레인 헬릭스 1 앞의 서열은 현저한 특징(distinguishing features)을 가지지 않았다.
트랜스멤브레인 헬릭스 1 은 모티프 [L*IVM]-Y*-[FL]-x(13)-[YF]-D*(SEQ ID NO: 1)을 포함한다.
트랜스멤브레인 헬릭스 3 은 현저한 특징을 가지지 않는다.
트랜스멤브레인 헬릭스 4 는 현저한 특징을 가지지 않는다.
트랜스멤브레인 헬릭스 6 은 모티프 W* R-[IVLA]-P-x(3)-Q (SEQ ID NO: 5)을 포함한다.
트랜스멤브레인 헬릭스 8 는 현저한 특징을 가지지 않는다.
트랜스멤브레인 헬릭스 9 는 현저한 특징을 가지지 않는다.
트랜스멤브레인 헬릭스 12 는 현저한 특징을 가지지 않는다.
트랜스멤브레인 헬릭스 12 뒤의 서열은 현저한 특징을 가지지 않는다.
NCU00801 및 NCU08114 의 상동 모델(Homology models)은, zhanglab.ccmb.med.umich.edu/I-TASSER/(Roy 등의, 2010)에서 I-TASSER 서버를 사용한 NCU00801 and NCU08114 의 일차아미노산 서열(primary amino acid sequences)로부터 생성되었다. I-TASSER 에 의해 생산된 최고의 구조적인 모델(top structural models)은 PYMOL(웹페이지 pymol.org/)에서 확인하였다. 모티프의 매핑(Mapping)을 PYMOL 에서 또한 실시하였다. 표시된 셀로덱스트린 운반체 모티프를 갖는 NCU00801 및 NCU08114 의 상동 모델(homology models)을 도 29(A,B)에 나타내었다. 도 29C 는 NCU00801 및 NCU08114 의 예측된 2 차 구조(secondary structures)를 나타낸 것이다.
사카로미세스
세레비시아에서
네우로스포라
크라사
및
피치아
스티피티스로
부터의
신규한
오탄당-특이적인 운반체의 특징
본 실시예에서, 생물정보학적 접근(bioinformatics approach)은 N. 크라사 및 P. 스티피티스에서 신규한 오탄당-특이적인 운반체를 확인하기 위해 사용되었다.
오탄당-특이적인 운반체의 게놈 발굴(
genome
mining
)
생물정보학적 연구
신규한 D-자일로스-특이적인 운반체를 발견하기 위해서, C. 인터메디아로부터의 D-글루코스/D-자일로스 공수송체 Gxsl(Leandro 등의, 2006) 및 P. 스티피티스로부터의 특징지어지지 않은 추정상의 L-아라비노스-양성자 공수송체(symporter) Autl[로커스 태그(locus tag) PICST_87108]를 코딩하는 유전자를 두 가지의 효율적인 자일로스-활용 종(xylose-utilizing species), N. 크라사 및 P. 스티피티스의 배열순서가 밝혀진 게놈(sequenced genomes)에 대해서 BLAST 검색(웹페이지 ncbi.nlm.nih.gov/)에서의 프로브(probes)로서 사용하였다(Galagan 등의, 2003; Jeffries 등의, 2007). 알려진 D-글루코스 운반체 활성도 또는 당 수송 외의 활성도를 갖는 모든 단백질은 분석에서 제거되었다. 25 % 최소의 서열 동일성의 차단(cut-off)을 사용하여, P. 스티피티스로부터의 AUT1 뿐만 아니라 17 가지의 추정상의 오탄당 운반체 유전자를 확인하였다(표 15). 이러한 추정상의 오탄당 운반체 유전자는 C. 인터메디아로부터의 GXS1 또는 P. 스티피티스로부터의 AUT1 와 20-25 % 동일성을 공유한다. 모든 17 가지 추정상의 오탄당 운반체는 당-수송 단백질 또는 알려지지 않는 활성도(unknown activity)를 갖는 가설 단백질(hypothetical proteins)로서 주석을 달았다(annotated). P. 스티피티스로부터의 D- 글루코스 운반체 유전자 SUT1 및 SUT2 는 비교를 위해 또한 클론하였다.
[표 15]
추정상의
오탄당 운반체의 클로닝(
cloning
)
N. 크라사 및 P. 스티피티스를 탄소원으로서 D-자일로스 또는 L-아라비노스 둘 중 하나로 보충된 리치 배지(rich media)에서 배양하였다(cultivated). 전체 RNA 를 분리하였고, cDNA 내로 역전사하였다(reverse transcribed). 중합효소 연쇄반응(PCR)은 cDNA 로부터 직접적으로 추정상의 운반체 유전자를 증폭시키는데 사용하였다. 그러나 균종(fungal species)에서의 오탄당 운반체의 조절 메카니즘 및 발현 패턴은 잘 알려지지 않았기 때문에, 배양 조건(cultivation conditions)에서 변경(alteration)에도 불구하고 추정상의 오탄당 운반체를 코딩하는 cDNA 를 항상 수득할 수 있는 것은 아니다. 이러한 경우에서, 프라이머를 GenBank 로부터의 이에 상응하는 cDNA 서열에 따라 설계하였고(designed), 주형(template)으로서 게놈의(genomic) DNA 를 갖는 엑손(exons)을 증폭시키는데 사용된다. 오버랩-연장 PCR(Overlap-extension PCR)은 전체 길이의 유전자(full-length genes) 내로 엑손을 모으는데 사용된다. 효모 상동의 재조합-매개 DNA 조립 방법을 사용한 pRS424-HXT7-GFP 셔틀벡터 내로 결과적으로 생성된 PCR 산물을 클론하였다(Shao 등의, 2009). 이러한 플라스미드에서, HXT7 프로모터, 둘 다의 말단에서의 EcoRI 사이트를 갖는 측면에 배치된 GFP 유전자 및 HXT7 종결자를 Cla1 및 BamHI 에 의해 선모양으로 만든(linearized) pRS424 셔틀벡터(New England Biolabs) 내로 조립하였다. HXT7 프로모터 및 종결자와 서열 동일성(sequence identity)을 공유하는 DNA 단편과 함께 측면 배치된 추정상의 오탄당 운반체의 PCR 산물(도 30A)을, 표준 리튬 아세테이트 방법을 사용한 pRS424-HXT7-GFP 소화된 EcoRI 를 갖는 Euroscarf (독일 프랑크푸르트에 위치한)로부터 입수한 S. 세레비시아 CEN.PK2-1C 스트레인(MATα leu2-3,112 ura3 -52, trpl -289, his3 -△l MAL2 -8c) 내로 공동으로 이동되었다(co-transferred). 결과적으로 생성된 혈질전환 혼합물(transformation mixture)을 2 % 글루코스로 보충된 SC-Trp 플레이트에 도말하였다(plated).
Zymoprep Yeast Plasmid Miniprep II(미국 캘리포니아 오렌지에 위치한 Zymo Research)를 사용한 형질전환체로부터 분리된 효모 플라즈마를 대장균 DH5a 세포 (미국 일리노이주 어배너에 위치한 일리노이드 대학의 어배너-샴페인, Cell Media Facility)내로 재-이동시켰다. 플라스미드를 QIAprep Spin Miniprep Kit(미국 캘리포니아 발렌시아에 위치한 QIAGEN)을 사용하여 분리하였고, 그리고 난 다음에 원본 운반체 유전자를 증폭시키는데 사용된 프라이머를 갖는 진단 PCR(diagnostic PCR)에 의해 체크하였다. 전체적인 오픈리딩프레임(entire open reading frames)을 정확한 구성(미국 일리노이드 어배너에 위치한 일리노이드 대학의 어배너-샴페인, Core Sequencing Facility)을 확인하기 위한 DNA 시퀀싱(DNA sequencing) 또한 제출하였다. DNA 시퀀싱 결과(DNA sequencing results)를 Sequencher 4.7(미국 미시간주 앤 아버에 위치한 Gene Codes Corporation)를 사용한 데이터베이스에서의 유전자 서열과 비교하였다. 클론된 추정상의 운반체의 모든 서열은 SEQ ID NOs: 33-52 에 작성하였다.
플라스미드를 유지하기 위해 합성 드롭아웃 배지(synthetic dropout media)에서 효모 스트레인을 배양하였다[0.17% 아미노산 및 황산암모늄이 없는 Difco 효모 질소 염기(yeast nitrogen base), 0.5% 황산암모늄, 0.05% 아미노산 드롭아웃 믹스(amino acid drop out mix)]. 2 % 의 당으로 보충된 YPA 배지를 플라스미드를 함유하지(harboring) 않는 효모 스트레인을 성장시키는데 사용하였다[1 % 효모 추출물, 2 % 펩톤, 0.01 % 아데닌 헤미술페이트(adenine hemisulfate)]. S. 세레비 시아 스트레인을 호기성 성장(aerobic growth)에서 30 ℃ 및 250 rpm 으로 배양시켰고, 산소-제한된 조건에서 30 ℃ 및 100 rpm 으로 배양시켰다. 효모 스트레인을 다른 방식으로 특정하지 않았으면 세포 조작(cell manipulation)을 위한 호기성 조건 하에서 성장시켰다. 대장균 스트레인을 Luria broth(LB)(미국 펜실베니아 피치버그에 위치한 Fisher Scientific)에서 37 ℃ 및 250 rpm 으로 배양하였다. 모든 제한 효소를 New England Biolabs(미국 매사추세츠 입스위치에 위치)로부터 입수하였다. 모든 화학물질을 Sigma Aldrich(미국 미주리주 세인트루이스에 위치) 또는 Fisher Scientific 로부터 입수하였다.
클론된
추정상의
운반체에 대한 운반체 활성도 검정
오탄당의 세포 내의 축척
클론된 추정상의 오탄당 운반체를 S. 세레비시아 당 운반체 결실 스트레인에서 과잉-발현되었고(over-expressed), 오탄당의 흡수를 측정하였다. 추정상의 오탄당 운반체의 D-자일로스-흡수 능력을 세포 내의 D-자일로스 및 자일리톨 농도의 요약(summation)으로 결정하였다. S. 세레비시아 세포 내에 축적된 D-자일로스가 내인성 알도스 환원효소(endogenous aldose reductase)의 존재로 인하여 부분적으로 자일리톨로 전환될 수 있다. D-자일로스 및 자일리톨 둘 다는 삼투현상을 사용하여 추출하였고, 고성능 액체 크로마토그래피(high performance liquid chromatography, HPLC)를 사용하여 분석하였다.
교수 E. Boles' laboratory 로부터의 선물(Institut fur Mikrobiologie, Heinrich-Heine-Universitat, Universitatsstr. 1, Geb. 26.12.01, D-40225 Diisseldorf, Germany)인 당 운반체 넉아웃(knock-out) S. 세레비시아 스트레인 EBY.VW4000(CEN.PK2-lc △hxtl-17, △stll, Aagtl, △ydl247w, △yjrl60c, △gal2)은 HXT1 -17 및 GAL2 를 포함하는 20 가지 보다 많은 당 운반체의 공존하는 넉아웃(concurrent knock-outs) 및 감지기(sensors)를 갖는다. 단일 탄소원으로서 D-글루코스에서의 성장은 이러한 스트레인에서 완전하게 중단된(abolished) 반면에, 상이한 당 수송 시스템(sugar transport system)을 통한 말토스의 흡수는 유지되었다. EBY.VW4000 스트레인은, 재조합 D-자일로스 흡수를 테스트하기 위해 이를 적절한 숙주로 만드는, HPLC 검정 조건 하에서 최소의 오탄당-흡수를 또한 나타내었다. 클론된 추정상의 오탄당 운반체 유전자가 과잉-발현된(over-expressing) 플라스미드를, 표준 리튬 아세테이트 방법을 사용한 EBY.VW4000 스트레인 내로 이동시켰고, 단일 콜로니(single colonies)를 당 흡수 활성도를 측정하기 위해 사용하였다.
세포를 2 % 말토스로 보충된 2 mL SC-Trp 배지에서 첫 번째로 배양하였다. 그리고 난 다음에 종균배양(Seed culture)을 250 mL 의 플라스크에 50 mL 배양물을 배양하는데 사용하였다. 세포를 24 시간 성장한 후에 원심분리하여 채취하였고, 10 의 최종 OD600 에 대한 2 % D-자일로스 또는 L-아라비노스로 보충된 YPA 배지에서 재-현탁시켰다(re-suspended). 30 분, 60 분, 120 분 및 24 시간에서, 5 mL 배양물을 세포내 당 농도를 측정하기 위해 가져왔다. 배양물 샘플을 얼음처럼 차가운 물로 2 번 세척하고 3 mL 의 탈이온수에서 재-현탁시켰다. 세포 현탁액을, 세포내의 당을 추출하기 위해 2 일 동안 37 ℃ 에서 250 rpm 으로 교반시키면서 배양하였다. 결과적으로 생성된 세포 현탁액을 HPLC 분석 전에 0.22 ㎛ PES 필터(미국 매사추세츠주 로웰에 위치한 Corning)를 통해 여과하였다. 당 및 이에 해당하는 당 알코올(하기에 기재된)의 농도를, 제조업자의 프로토콜(manufacturer's protocol)에 따른 Shimadzu ELSD-LTII 저온도-증발에 의한 광산란 검출기(low temperature-evaporative light scattering detector)(Shimadzu) 및 BioRad HPX-87C 컬럼(미국 캘리포니아주 허큘러스에 위치한 BioRad Laboratories)이 갖추어진 Shimadzu HPLC 을 사용하여 결정하였다. 당-흡수 활성도를 OD ~ 10 에서, 세포 배양물의 mL 에 대한 삼투압을 통해 추출된 당의 mg 으로서 계산하였다.
몇몇의 추정상의 오탄당 운반체는, D-글루코스 또는 D-자일로스, 또는 둘 다의 흡수에서 유효한 것을 확인되었다. D-글루코스는 효모 내에서 한번 대사작용을 할 수 있기 때문에(metabolized), D-글루코스 수송 활성도는 세포내의 D-글루코스 농도를 측정하여 결정할 수 없다. 그러나, EBY.VW4000 스트레인은 단일 탄소원으로서 D-글루코스를 포함하는 배지에서 성장할 수 없기 때문에, D-글루코스에서 추정상의 오탄당 운반체로 형질전환된(transformed) 스트레인의 성장은, 추정상의 오탄당 운반체가 D-글루코스 수송 활성도를 갖음을 나타낸다.
SUT3(Xyp37), XUT3(Xyp33), SUT2(Ap31), NCU04963(An29-2) 및 NCU06138(Xy31)의 도입(Introduction)은 D-글루코스에서 EBY.VW4000 스트레인의 성장을 회복시키고(restored), 따라서 글루코스 수송 활성을 가능하게 한다. SUT3, XUT3, SUT2 및 NCU04963 은 자일로스 수송 활성도를 또한 가지는 반면에, NCU04963 및 NCU06138 은 아라비노스 수송 활성도를 나타내었다(도 31). 추정상의 운반체의 나머지는 D-글루코스에서 성장을 가능하지 못하게 하고, 이들 중 대부분은 어떠한 오탄당 수송 활성도를 나타내지 않았다. 그러나 NCU00821 및 STL12/XUT6 은 자일로스 수송 활성도를 나타내지만, XUT1 은 아라비노스 수송 활성도를 나타내었고, 이는 이들인 오탄당에 대해 특이적인 당 운반체임을 나타낸다(도 32).
P. 스티피티스로부터의 STL12/XUT6 및 XUT1, 및 N. 크라사로부터의 NCU00821 가 D-글루코스-흡수 활성도를 갖지 않는 실제로 오탄당-특이적인 운반체임을 추가적으로 확인하기 위해, 당-흡수 검정을, 기질로서 14C-표지된 D-글루코스, D-자일로스 및 L-아라비노스를 사용하여 실행하였다. STL12/XUT6 및 NCU00821 만이 과잉-발현된(over-expressing) EBY.VW4000 스트레인의 D-글루코스- 및 L-아라비노스-흡수 활성도는, 둘 다의 운반체의 D-자일로스-흡수 반응속도를 측정하기 위해 사용된 검정 조건 하에서 측정에 대해서 매우 낮은 것으로 발견되었다.
14C-표지된 D-글루코스, L-아라비노스 및 D-자일로스를 90 % 에탄올에서의 용액으로서 American Radiolabeled Chemicals(미국 미주리주 세인트루이스에 위치)로부터 입수하였다. 방사능 표지된 당(Radiolabeled sugars)을 화학적 후드(chemical hood)에서 첫 번째로 건조시키고 난 다음에 물에 재-현탁시켰다. 대략 40,000 dpm/μL 의 특이적인 방사능(radioactivity)을 갖는 1.33 M 및 1 M 의 농도 및 약 20,000 dpm/μL 의 특이적인 방사능(radioactivity)을 갖는 500 mM, 350 mM, 250 mM, 100 mM 및 50 mM 의 농도의 당 용액(Sugar solutions)을 당-흡수 검정에서 사용하였다. 지수기(exponential phase)에서 세포 배양물을 채취하였고, 얼음처럼 차가운 물로 두 번 세척하고, pH 5 의 100 mM 트리스-시트레이트 완충용액(Tris-citrate buffer)에 대해 mL 당 약 60 mg 의 세포 건조무(dry cell weight, DCW)에 재현탁시켰다. 160 μL 의 세포 현탁액의 세 개의 앨리쿼트(aliquots)를 DCW 를 측정하기 위해 24 시간 동안 65 ℃ 에서 건조시켰다. 세포 현탁액의 나머지를 사용하기 전에 얼음에 보관하였다. 당-흡수 검정에 대해서, 세포 현탁액을 검정하기 전에 5 분 동안 30 ℃ 에서 평형을 유지시켰다. 50 mL 의 원추형 튜브(conical tube)에서, 160 μL 의 세포 현탁액을 40 또는 60 초(정확한 시간) 동안 방사능-표지된 당 용액(radio-labeled sugar solution)을 혼합하였다. 주사기(syringe)로 운반된(delivered) 얼음처럼 차가운 10 mL 의 물을 첨가하여 반응을 정지시켰다. 제로 시점 샘플(zero-time-point sample)을 얼음처럼 차가운 물 및 방사능-표지된 용액을 함유하는 배양 튜브(culture tube)에 동시에 첨가하여 수득하였다. 혼합물을, 40 % 의 당 용액에 미리-흠뻑 젖은(pre-soaked) Whatman GF/C 필터(미국 뉴저지 플로함 파크에 위치한 Whatman)를 통해 즉시 여과하였고, 15 mL 의 얼음처럼 차가운 물(ice-cold water)로 세척하였다. 필터를 3 mL 의 Econo I 섬광 칵테일(scintillation cocktail)(Fisher Scientific)에 두었고, 1 분 동안 Beckman LS6500 신틸레이션 계수기(scintillion counter)(미국 캘리포니아주 브레아에 위치한 Beckman Coulter)를 사용하여 계수하였다. 모든 측정점(data points)는 세 가지의 독립적인 실험으로 측정하였다. 당-흡수 비율을 mmol 의 수송된 당/시간/그램의 세포건조무(dry cell weight)로 계산하였다.
STL12/XUT6, NCU00821 또는 XUT1 이 과잉-발현된 EBY.VW4000 스트레인에서 D-자일로스 및 L-아라비노스 둘 다의 세포내 축적(Intracellular accumulation)은 HPLC 를 사용하여 또한 측정하였다. 30 분, 60 분, 120 분 및 24 시간 동안의 오탄당 당(pentose sugars)과 함께 배양한 세포 배양물을 HPLC 로 분석하였다. STL12/XUT6 또는 NCU00821 가 과잉-발현된 EBY.VW4000 스트레인은 D-자일로스 흡수 활성도를 나타낸 반면에, XUT1 과잉-발현된 스트레인은 24 시간 배양 후에 L-아라비노스-흡수 활성도를 나타내었다(도 33).
세포내 당 축척의 HPLC 분석을 포함하여 14C-표지된 당 흡수 검정(sugar uptake assay)은, 리그노셀룰로직계 가수분해물(lignocellulosic hydrolysates), D-글루코스, D-자일로스 및 L-아라비노스에서 세 가지 가장 풍부한 단당류 중에서, STL12/XUT6 및 NCU00821 은 D-자일로스 흡수에 대한 책임이 있고, XUT1 은 L-아라비노스 흡수에 대해 책임이 있다. 중요한 것은, D-자일로스 흡수에 대한 효모에서 연구된 대부분 당 운반체는 D-자일로스 보다는 D-글루코스에 대해 보다 높은 흡수 활성도를 갖는다. 적응진화(adaptive evolution) 후에 트리코데르마 레세이로부터의 Trxltl 만이 D-자일로스-특이적인 흡수 활성도를 나타내었다(Saloheimo 등의, 2007). 이러한 데이타는, P. 스티피티스로부터의 STL12/XUT6 및 N. 크라사로부터의 NCU00821 는 S. 세레비시아 내로 도입된(introduced) 첫 번째 두 가지의 실험적으로 확인된 자연 발생적으로(naturally)-발생한(occurring) D-자일로스-특이적인 운반체임을 나타낸다. 마찬가지로, P. 스티피티스로부터의 XUT1 은, S. 세레비시아 내로 도입된(introduced) 첫 번째 실험적으로 확인된 자연 발생적으로(naturally)-발생한(occurring) L-아라비노스-특이적인 운반체임을 나타낸다.
동역학적 파라미터(
Kinetic
parameters
)
14C-표지된 당-흡수 검정을 사용하여, NCU00921, STL12/XUT6 및 XUT1 를 통한 D-자일로스 수송의 동역학적 파라미터를 확인하였다. 검정 조건 하에서, 당 흡수는 첫 번째 60 초 동안의 직선 범위(linear range) 내임이 관찰되었다(도 34). NCU00821, STL12/XUT6 또는 XUT1 가 과잉-발현된 EBY.VW4000 스트레인은 40 또는 60 초 동안 표지된 D-자일로스 또는 L-아라비노스와 함께 배양한 다음에, 추가적인 당 흡수를 정지시키기 위해 얼음처럼 차가운 물을 첨가하였다. 액체 섬광 계수기(liquid scintillation counter)를 사용하여 측정하기 전에 반응 혼합물을 여과한 다음에, 세척하였다. 당- 흡수 비율 및 기질 농도를, Origin 스프트웨어(미국 매사추세츠주 노샘프턴에 위치한 OriginLab Corporation)를 사용한 비선형회귀(non-linear regression)에 의한 미하엘리스-멘텐 방정식(Michaelis-Menten equation)에 적용시켰다(fitted). NCU00821 또는 STL12/XUT6 만을 포함하는(harboring) EBY.VW4000 스트레인에 의해 D-자일로스 흡수에 대한 K m 값은 각각 175.7 ± 21.4 mM 및 56.0 ± 9.4 mM 이다. 이에 해당하는 V max 값은 각각 36.7 ± 2.9 및 41.5 ± 2.3 μmol/h/gram DCW 이다. 유사하게, XUT1 을 포함하는 EBY.VW4000 스트레인에 의한 L-아라비노스 흡수에 대한 K m 및 V max 값은 각각 48.0 ± 13.2 mM 및 5.6 ± 1.6 μmol/h/gram DCW 이다.
자연 발생적으로-발생한 D-자일로스-동화 균종(assimilating fungal species)에서의, 높은 친화도의 D-자일로스-양성자 공동수송 시스템(symport system) 및 낮은 친화도의 D-자일로스 촉진 확산 시스템(facilitated diffusion system)이 존재한다. 이러한 두 가지 시스템의 K m 값은 공동 수송 시스템에 대한 0.4-4 mM 및 촉진 확산 시스템에 대한 약 140 mM 인 것으로 결정되었다(Leandro 등의, 2006 ; Stambuk 등의, 2003). 이러한 값은 S. 세레비시아에서 D-글루코스-흡수 시스템의 친화도와 가깝고(close), 이는 높은 친화도 시스템에 대한 1.5 mM 의 K m 및 낮은 친화도 시스템에 대한 20 mM 의 K m 을 갖는다(Lang and Cirillo 1987 ; Ramos 등, 1988). 불행하게도, 야생형 S. 세레비시아의 D-자일로스 흡수 친화도는 D-글루코스에 대한 이의 친화도 보다는 10 의 두 제곱(two orders of magnitude)이나 낮았다. S. 세레비시아에서 D-자일로스 흡수에 대한 K m 값은 높은 친화도 시스템에 대해 오직 190 mM 이고, 낮은 친화도에 대해 1.5 M 이다(Kotter and Ciriacy, 1993). 자연 발생적으로 발생한 D-자일로스-동화(assimilating) 효모에서 높은 친화도의 D-자일로스-흡수 시스템과 비교하였을 때, 새로이 발견된 D-자일로스-특이적인 운반체의 친화도는 보다 낮았다. 그러나, 야생형 S. 세레비시아에서 D-자일로스-흡수 시스템과 비교하였을 때, NCU00821 및 STL12/XUT6 은 D-자일로스에 대한 보다 높은 친화도를 나타내었다. 특히, STL12/XUT6 및 XUT1 에 의한 D-자일로스의 K m 은 야생형 S. 세레비시아에서 운반체에 의한 자일로스 흡수 K m 의 오직 4 분의 1 (one- fourth of K m )이다. D-자일로스-특이적인 운반체의 K m 값은 Gxfl(K m 88 mM) 및 Sutl(K m 145 mM)의 이들에 가깝고, 이는 재조합 S. 세레비시아에서 D-자일로스 발효를 증진시키는 것으로 나타났다(Runquist 등의, 2009 ; Katahira 등의, 2008). 따라서, D-자일로스 발효는, S. 세레비시아 내에 이러한 최근에 발견된 D-자일로스-특이적인 운반체를 도입하여 증진시킬 수 있다.
당 운반체의
세포내
위치(
cellular
localization
)
당 운반체는 트랜스멤브레인 단백질이고, 세포막(cell membrane)에서의 정확한 폴딩(correct folding) 및 위치(localization)는 기능적일 수 있는 이들을 필요로 한다. 추정상의 오탄당 운반체가 클론되었을 때, 신호 펩티드가 특이적으로 첨가되지 않았기 때문에, D-자일로스-특이적인 운반체가 세포막(cell membrane)에 정확하게 위치하는 것을 보장하기 위해 이는 중요하다. 이는 곰팡이 N. 크라사로부터 클론된 추정상의 오탄당 운반체 유사 NCU00821 에 대해서 특히 적용되었고(true), 이는 S. 세레비시아와 비교한 매우 상이한 생리(physiology)를 나타낸다. S. 세레 비시아에서의 D-자일로스-특이적인 운반체의 세포내 위치를 연구하기 위해, NCU00821, STL12/XUT6 및 XUT1 을 링커(linkers)를 통해 C-말단에서 녹색 형광 단백질(Green Fluorescent Protein, GFP)로 융합하였고, 이들의 위치(their localization)를 형광 영상(fluorescent imaging)으로 모니터하였다.
C-말단에서 GFP 를 갖는 오탄당-특이적인 운반체의 융합 단백질(fusion proteins)은 운반체 위치 연구(transporter localization study)를 위해 구성하였다. GS-링커[Gly-Gly-Gly-Gly-Ser-Gly-Gly-Gly-Gly- Ser (SEQ ID NO: 70)]를 운반체 및 GFP 사이에 도입하였다. GS-링커를 PCR 프라이머에 의한 GFP 오픈 리딩 프레임(open reading frame)의 N-말단에 첨가하였고, 이로 인하여 5'-말단에서의 운반체 및 3'-말단에서의 HXT7 종결자(terminator)에 상동인 뉴클레오티드 서열을 측면에 둔(flanked with) GS-링커-GFP 의 PCR 산물이 생성되었다. 운반체 유전자는, 5'-말단에서의 HXT7 프로모터 및 3'-말단에서의 GS-링커-GFP 와 동일한 뉴클레오티드 서열을 측면에 둔(flanked with) 운반체의 DNA 단편을 생성하기 위해 원본의 pRS424-HXT7-운반체 구조물(constructs)로부터 증폭시켰다. 그리고 난 다음에 이러한 두 가지 단편을, EcoRl 로 소화된 (digested) pRS424-HXT7-GFP 로 S. 세레비시아 스트레인 CEN.PK2-1C 내로 공동-이동시켰다(co-transferred)(도 30B). 결과적으로 생성된 형질전환 혼합물을 2 % D-글루코스로 보충된 SC-Trp 플레이트에 도말하였다(plated).
단일 콜로니를 2 % 말토스로 보충된 2 mL 의 SC-Trp 액체 배지 내로 접종하였다(inoculated). 세포 배양물을 지수기(exponential phase)에서 채취하였다. 원심관(centrifuge tube)에서, 250 μL 의 세포 배양물을 10 분 동안 실온에서 10 μL 의 Hoechst 33342 핵 염료(nuclei dye)(미국 캘리포니아주 칼즈배드에 위치한 Invitrogen)로 염색하였다(stained). 세포 배양물의 소량의 물방울(small droplet)을 한 장의 커버 글라스에 이동시킨 다음에, 형광 이미지(fluorescent images)를 Andor Technology Revolution System 회전원판식 공초점 현미경(Spinning Disk Confocal Microscope)(미국 일리노이주 어배너 일리노이대학교 어배너-섐페인의 Institute for Genomic Biology 에 위치한 Core facilities)을 사용하여 찍었다. 이미지를 Imaris 화상해석(image analysis) 및 시각화 소프트웨어(visualization software)(미국 미네소타주 세인트 폴에 위치한 Bitplane)을 사용하여 진행하였다.
오탄당-특이적인 운반체가 과잉-발현된(over-expressing) 효모 스트레인을 세포 주변부(cell periphery)에서 독특한 형광 헤일로(fluorescent halo)를 나타내었다(도 35). NCU00821 및 XUT1 에 대해서, 대부분의 모든 GFP 형광은 세포막에서 나타난 반면에, STL12/XUT6-과잉-발현된 세포에서의 형광의 대부분은 세포질(cytoplasm)에 잔존하였다. 이는 막 단백질(membrane protein)의 상승된 발현 때문에 STL12/XUT6 운반체의 비효율적인 유출(export)을 나타낸다. 모든 세포가 형광을 나타내지 않았고 이는 운반체의 발현이 최적이지 않음을 나타내는 점에 또한 주목하였다. 운반체 발현의 추가적인 개선은 발현 수준의 변경 및/또는 재조합 S. 세레비시아의 게놈 내로 운반체 유전자의 통합을 통해 성취될 수 있다.
오탄당 운반체의 유형의 확인(
Determination
)
S. 세레비시아에서의 당 운반체의 두 가지 유형, 공수송체(symporters) 및 촉진제(facilitators)가 있다. 공수송체에 대해서, 당 흡수는 양성자 흡수와 결합되어있다. 당 공수송체는 당에 대해서 일반적으로 높은 친화도를 나타내었다. 그 동안에, 촉진제를 통한 당 흡수는 양성자와 결합되지 않았지만, 촉진제는 낮은 당-흡수 친화도를 일반적으로 나타내었다(Leandro et al., 2006). 공수송체 검정은 EBY.VW4000 스트레인에서 발현된 NCU00821, STL12/XUT6 및 XUT1 에 대해서 실시하였다.
운반체의 유형을 결정하기 위해, EBY.VW4000 과잉-발현된 오탄당-특이적인 운반체의 pH 변화를, USB 통신장치(communication module) 및 Direct pH 소프트웨어(미국 오하이오주 콜럼버스에 위치한 Mettler Toledo)가 갖추어진 Seven Multi pH 미터(meter)를 사용한 D-자일로스, L-아라비노스 또는 말토스를 포함하는 완충되지 않는(un-buffered) 세포 현탁액에서 측정하였다. 오탄당-특이적인 운반체를 코딩하는 플라스미드를 EBY.VW4000 스트레인 내로 이동한 후에, 2 % 말토스로 보충된 SC-Trp 플레이트에 도말하였다. 단일 콜로니를 2 % 말토스로 보충된 2 mL SC-Trp 배지에서 배양하였다. 2 L 의 플라스크에 400 mL 배양물을 접종하기 위해 종균 배양(Seed culture)을 사용하였다. 배양물을 OD ~ 1 에서 채취하였고, 얼음처럼 차가운 물(ice-cold water)에 두 번 세척하였다. 세포 펠릿(Cell pellets)을 4 mL 의 물에 재-현탁시키고 사용하기 전에 얼음에 두었다. 공수송체 검정을 위해, pH 전극을 25 ℃ 에서 유지시킨 50 mL 용량(capacity)의 물-자켓 비커(water-jacketed beaker)에 담그었고, 마그넨틱 교반(magnetic stirring)을 제공하였다. 비커에, 25 ℃ 에서 평형시킨(equilibrated) 23 mL 의 탈이온수(deionized water) 및 1 mL 의 세포 현탁액을 첨가하였다. pH 를 5 에 조정하였고, 베이스라인(base line)을 획득하였다. pH 변화를 pH 5 에서 1 mL 의 50 % 당 용액을 첨가하여 기록하였다.
도 36 은 말토스의 첨가 후에 완충되지 않은(un-buffered) 세포 현탁액에서의 pH 변화를 나타낸 것이다. 기록된 바와 같이, 완충되지 않은 S. 세레비시아 세포 현탁액에서 pH 는 말토스를 첨가하면서 상승되었다. pH 기록 시스템(pH recording system)이 가동되는 것을 확인하기 위해, 1 mL 의 50 % 말토스 용액을 첨가하였다. 모든 샘플에서 관찰된 pH 증가(elevation)는, pH 기록 시스템이 실험적인 세팅(experimental setting)에서 일시적인 pH 변화를 모니터할 수 있음을 나타낸다.
완충되지 않은 세포 현택액에서 pH 증가가 없는 것은 오탄당-특이적인 운반체의 어떠한 것에 대해서 관찰되었고, 이는 이러한 운반체를 통한 오탄당 흡수가 양성자 수송과 결합되어 있지 않음을 나타낸다(도 37). 따라서, NCU00821, STL12/XUT6 및 XUT1 을 오탄당 촉진제일 수 있는 것으로 결정되었다.
이러한 결과는, NCU00821 및 STL12/XUT6 의 동역학적 파라미터는 자연적으로-발생한 D-자일로스-동화 효모(assimilating yeasts)에서 낮은 친화도 D-자일로스를 가능하게 하는 확산 시스템(low affinity D-xylose facilitated diffusion system)의 이러한 것과 유사한 사실과 일관된다. 공수송체는 D-자일로스에 대해 보다 높은 친화도를 갖는 사실에도 불구하고, 공수송체의 과잉-발현은, 양성자 구배(proton gradient)를 생성하기 위한 ATP 필요성 때문에 D-자일로스-동화 스트레인에 의한 당 이용을 항상 용이하게 할 수 있는 것은 아니다. 사실은, D-자일로스 발효에 유익한 것으로 나타낸 대부분의 운반체는 촉진제이다(Runquist et al., 2009; Katahira el al., 2008).
D-
자일로스
-특이적인 운반체의 이종 기원 과잉-발현(
Heterologous
Over
-
Expression
)
D-자일로스 이용 경로를 포함하는 S. 세레비시아 스트레인에서 활성(active) 이종 기원 D-자일로스-특이적인 운반체의 과잉-발현은, 이들의 과잉-발현이 자일로스 이용을 개선시키는지 여부를 확인하기 위해 또한 조사하였다. 자일로스 이용은 호기성 조건 하에서 쉐이크플라스크(shake-flask)를 사용하여 연구하였다. 자일로스 운반체 NCU00821, NCU04963, XUT1, STL12/XUT6 및 Hxt7 이 발현된 플라스미드를, 스트레인 HZE63 내로 도입하였다[CEN.PK2 ura3::자일로스 이용 경로(xylose utilization pathway)]. 이러한 스트레인은 염색체 내로 URA3 부위(site) 내로 통합된 자일로스 이용 경로를 갖는다. 이는, N. 크라사로부터 자일루로스 환원효소(xylulose reductase, XR) 및 자일리톨 탈수소효소(xylitol dehydrogenase, XDH) 및 P. 스티피티스로부터 자일루로키나아제(XKS)를 포함하는, 이전의 작업으로부터 플라스미드를 사용하여 구성하였다. 스트레인 HZE63 를 수득하기 위해 이러한 플라스미드를 ApaI 로 절단하고, 효모 스트레인 CEN.PK2 내로 형질전환시켰다(transformed).
자일로스 운반체-코딩 플라스미드로 형질전환시킨(transformed) HZE63 스트레인을 2 % 글루코스로 보충된 SC-Ura 플레이트에 도말하여 선별하였다. 변형된 스트레인을 2 % 글루코스를 갖는 SC-Trp-Ura 에 예비-배양한 다음에, 초기의 OD600 = 1.0 에 대한 0.5 % 또는 5 % 의 자일로스로 보충된 SC-Trp-Ura 내에 접종하였다. 세포 배양물을, 250 rpm 으로 30 ℃ 에서 50 mL 의 배양물을 포함하는 125 mL 진탕-플라스크(shake-flask)에서 배양하였다(도 38).
형질전환체(transformant)의 효모 플라스미드를 E.coli DH5α 세포 내로 형질전환시켰다(transformed). 정확한 구조물(correct construction)을 확인하기 위해 플라스미드를 분리한 다음에, 진단 PCR(diagnostic PCR)로 체크하였고, 시퀀싱(sequencing)을 위해 제출하였다. 플라스미드 맵(Plasmid maps)을 도 39 에서 확인할 수 있다.
불행하게도, 오탄당-특이적인 운반체의 과잉-발현의 장점은 발현 전략의 변경, 배양 조건의 변경 및 D-자일로스 이용의 선택에도 불구하고 관찰할 수 없었다. 몇 가지 가능한 이유가 있었다. 첫 번째로, 당 운반체와 같은 막 단백질의 과잉-발현은 세포 막의 온전함(integrity)에 영향을 미칠 수 있고, 그 결과에 따라 세포의 성장을 방해할 수 있다(Wagner et al., 2006). D-글루코스를 탄소원으로서 사용하였을 때, 운반체 과잉-발현 스트레인은 보다 느린 성장 비율을 나타내는 것으로 관찰되었다. 글루코스-함유 SC-ura 배지에서 성장한 운반체를 전달하는 스트레인의 2-일 배양물의 최종 OD 는 오직 4 인 반면에, 음성 대조군(negative control)의 OD 는 약 6 이다. 두 번째로, 육탄당 운반체(hexose transporters)를 통한 야생형 S. 세레비시아의 D-자일로스-흡수 활성도는, 육탄당 운반체 녹아웃(knockout) 스트레인에서 과잉-발현된 특정 D-자일로스 운반체의 D-자일로스-흡수 활성도보다, 보다 높았다. 최근에 발견된 D-자일로스-특이적인 운반체의 낮은 당 수송 활성도는 당 흡수 능력의 개선을 관찰하기 어렵게 만들고 있다. 세 번째로, 신규한 D-자일로스-특이적인 운반체의 도입이 S. 세레비시아 세포 내로 D-자일로스의 흡수를 증가시킴에도 불구하고, D-자일로스 이용 경로가 당-흡수 제한 단계(sugar-uptake the limiting step)를 만들기에 충분하게 효율적일 때, D-자일로스 이용의 효과를 또한 관찰할 수 있다. 이는, 당 운반체의 과잉-발현의 효과가 스트레인의 백그라운드(strain background) 및 배양 조건에 의존하는 것으로 나타났다(Runquist et al., 2010). 하기의 실시예 12-15 는 효모에서 자일로스 이용 경로의 최적화(optimization)를 기재하였다.
추가적인 오탄당-특이적인 운반체의
클로닝
NCU00821, STL12/XUT6 및 XUT1 의 오솔로그를 클로닝하고(cloned) 오탄당 흡수를 위해 테스트하였다. 상이한 균류 스트레인(fungal strains)을 글루코스 또는 오탄당으로 보충한 리치 배지(rich media)에서 배양하였다. 전체 RNA 를 분리하였고, cDNA 내로 역전사되었다. 중합효소 연쇄 반응(Polymerase chain reaction, PCR)을 cDNA 로부터 직접적으로 추정상의 운반체 유전자(putative transporter genes)를 증폭시키는데 사용하였다. 그러나, 조절 매커니즘(regulation mechanism) 및 발현 패턴(expression pattern)이 균종(fungal species)에서의 오탄당 운반체에 대해서 알려지지 않았기 때문에, 추정상의 오탄당 운반체를 코딩하는 cDNAs 는 배양 조건의 변경에도 불구하고 항상 수득가능하지 않다. 이러한 경우에, 프라이머는 GenBank 로부터 이에 상응하는 cDNA 서열에 따라 설계되었고, 주형(template)으로서 게놈 DNA 를 사용한 엑손을 증폭시키기 위해 사용하였다. 오버랩-연장 PCR(Overlap-extension PCR)을 사용하여 전장 유전자(full-length genes) 내로 엑손을 조립하였다. 결과적으로 생성된 PCR 산물을 DNA 조립 방법(DNA assembler method)을 사용하여 HXT7 프로모터 및 HXT7 종결자(terminator)을 포함하는 pRS424 셔틀 벡터 내로 클론하였다. 형질전환체(transformants)로부터 분리된 효모 플라스미드를 E. coli DH5a 내로 재형질전환시키고(retransformed), 분리된 E. coli 플라스미드를 원본 운반체 유전자(original transporter genes)를 증폭시키는데 사용하는 프라이머를 사용하여 진단 PCR 로 처음으로 체크하였다. 전체 오픈 리딩 프레임(entire open reading frames)을, 플라스미드의 정확한 구조(correct construction)를 확인하기 위해 시퀀싱(sequencing)을 위해 제출하였다.
대부분의 클로닝 작업을 효소 이종 기원 재조합 매개 DNA 조립 방법(yeast homologous recombination mediated DNA assembler method)을 사용하여 실시하였다. pRS424-HXT7-GFP 플라스미드를 추정상의 오탄당 운반체의 클로닝(cloning)을 위해 사용하였다. 이러한 플라스미드에서, HXT7 프로모터, 두 말단에 EcoRI 사이트를 측면에 둔 GFP 유전자 및 HXT7 말단을, Clal 및 BamHI 에 의해 선모양으로 만든 pRS424 셔틀 벡터(New England Biolabs) 내로 조립하였다. HXT7 프로모터 및 종결자에 대해 서열 동일성을 공유하는(sharing) DNA 단편을 측면에 둔 추정상의 오탄당 운반체의 PCR 산물은, 표준 리튬 아세테이트 방법(standard lithium acetate method)을 사용하여 pRS424-HXT7-GFP 로 절단한(digested) EcoRI 로 CEN.PK2-1C 내로 공동-이동시켰다(co-transferred). 결과적으로 생성된 형질전환(transformation) 혼합물을 2 % D-글루코스로 보충된 SC-Trp 플레이트에 도말하였다(plated). 형질전환체를 오탄당 수송 활성도에 대해서 테스트하였다.
결과를 도 40 및 표 16 에 나타내었다. 8 가지의 추정상의 오탄당 특이적인 운반체[XP_960000 (NC52), CAG88709 (DH48), XP_457508 (DH61), XP_681669 (32- 10), XP_001487429 (29-6), XP_001727326 (29-9), XP_657854 (32-8), XP_720384 (29-4)] 중에서, NC52 만이 글루코스 플레이트에서 세포 성장할 수 있었고, 이는 그 밖의 7 가지 운반체가 오탄당-특이적이거나 불활성일수 있음을 암시한다. HPLC-기반 오탄당 흡수 검정을 사용하여, XP_457508(DH61), XP_001727326(29-9), XP_720384(29-4) 및 XP_681669(32-10)를 포함하는 4 가지 자일로스-특이적인 운반체를 발견하였다. 게다가, 하나의 아라비노스-특이적인 운반체, XP_657854 (32-8)를 확인하였다(도 40 ; Top). 5 가지의 추가적인 추정상의 오탄당 특이적인 운반체(XP_002488227, AB070824.1, XP_001389300, XP_002488227, EEQ43601.1)를 또한 테스트하였고, 이들 중 글루코스 플레이트에서 세포 성장할 수 있는 것은 없었다. 추가적인 오탄당 흡수 검정은, XP_002488227 및 AB070824.1 이 자일로스 특이적인 운반체임을 나타낸다(도 40 ; Bottom). 이러한 결과의 요약을 표 16D 에 나타내었다.
[표 16]
데이타베이스에서 서열과 불일치하는 서열을 갖는 오솔로그(예를 들어, 돌연변이를 갖는 것)을 재-클론하였고, 배열순서를 밝히고(sequenced), 효모 스트레인에서 발현시키고, 당 흡수 기능에 대해서 테스트하였다. 유사하게, 시퀀싱(sequencing) 결과가 없는 것에 대한 오솔로그 또한 운반체 기능에 대해서 테스트할 것이다.
오탄당 운반체 오솔로그의 서열 정렬을 보존 잔기를 확인하기 위해 분석하였고, 이는 운반체 기능에서 잠재적인 역할을 할 수 있다. 자일로스 운반체(NCU0821, STL12/XUT6, XP_002488227.1 및 XP_002382573.1) 및 아라비노스 운반체(XUT1 및 EEQ43601.1)의 샘플의 정렬을 도 41(A, B)에 각각 나타내었다. 몇몇의 잔기는 자일로스 운반체에서 특이적으로 보존된 반면에, 다른 것은 아라비노스 운반체에서 특이적으로 보존되었다. 이러한 잔기는 특이적인 오탄당을 수송하는데 중요한 역할을 가질 것이다. 자일로스 및 아라비노스 운반체의 서열의 전체적인 비교(도 41C)는, 둘 다의 유형의 오탄당 운반체에서 보존된 잔기가 있음을 나타내고, 이는 일반적인 오탄당의 흡수에서 기능적인 역할을 한다.
실시예 12-15 는 효모에서 자일로스 활용 경로의 최적화와 관련된 것이다.
오탄당-이용 S.
세레비시아
스트레인의 유전자 조작(
Engineering
)
효율적인 자일로스 대사경로는 동종효소(isoenzymes)의 개념을 이용하여 재구성하였다(reconstituted). 동종효소는 상이한 운동 특성 또는 조절 특성을 갖는 동일한 화학적 반응을 촉진시키고(catalyze), 세포질 환경(cytosolic environment)에서 동적인 변화(dynamic changes)에 대응하여 물질대사의 흐름(metabolic fluxes)의 미세-조정 조절(fine-tuned control)을 부여하기 위한 것으로 알려져있다. 그러나, 사전의 물질대사의 유전자 조작된 접근(metabolic engineering approaches) 없이, 관심을 갖는 흐름(fluxes of interest)을 증가시키기 위해 동종효소를 사용하였다. 이러한 연구는 야생형 및 돌연변이 자일루로스 환원효소(XR) 동종효소 둘 다의 동시의 발현은 자일리톨 축적을 감소시킬 수 있고, 전체적인 자일로스 발효 비율을 증가시킬 수 있음을 나타낸다.
살아있는 시스템(living systems)에서 동종효소의 출현율(prevalence)에 의한 영감에 의해, S. 세레비시아에서 기능적인 자일로스 대사 경로를 구성하기 위해, 자일리톨 탈수소효소(XDH) 및 자일루로키나아제(XR)와 함께 S. 세레비시아에서 야생형 XR 및 돌연변이체 XR(R276H)을 공동-발현시켰다(co-expressed). XR 돌연변이체는 NADH 상에서 NADPH 에 대해 보다 낮은 선호도(preference)를 나타낸 반면에, 야생형 XR 은 NADH 상에서 NADPH 에 대해 116 2-배(two-fold) 보다 높은 선호도를 나타내는 것이 보고되어 있다(Watanabe 등의, 2007).
P. 스티피티스로부터의 자일로스-대사작용하는 유전자(야생형 XYL1 , 2, 및 3 및 돌연변이체 XYL1)를, 발현 카세트(expression cassettes)를 구성하기 위해 PCR-증폭시키고, 구성요소 프로모토(constitutive promoters)(PGK1 and TDH3)의 조절 하에 두었다. 이러한 통합 카세트(integration cassettes)를 D452-2 스트레인의 게놈 내로 통합시켰다(integrated).
자일로스 대사 경로의 구성에 대한 발현 카세트의 형질전환(Transformation)은 효모 EZ-형질전환 키트(미국 캘리포니아주 비스타에 위치한 BIO 101)를 사용하여 실시하였다. 아미노산 영양요구 마커(auxotrophic marker)를 사용한 형질전환체(transformants)를 선별하기 위해, 효모 합성 완전한 배지[yeast synthetic complete (YSC) medium]를 사용하였고, 이는 6.7 g/리터(liter) 효모 질소 염기에 20 g/리터 글루코스, 20 g/리터 한천(agar) 및 CSM-Leu-Trp-Ura(BIO 101)을 더한 것을 함유하고, 이는 적절한 뉴클레오티드 및 아미노산을 공급한다. 효모 스트레인은 20 g/리터 글루코스를 갖는 YP 배지 234[10 g/리터 효모 추출물, 10 g/리터의 박토 펩톤(Bacto peptone)]에서 30 ℃ 로 일상적으로 배양하였다.
자일로스-대사작용 효율(xylose-metabolizing efficiency)에서의 S. 세레비 시아 스트레인 백그라운드(background)의 효과는 몇몇의 상이한 효모 스트레인에서의 최적화된 자일로스 활용 경로 효소를 함유하는 동일한 구조물의 발현에 의해 또한 테스트되었다. 사용된 3 가지 실험실 스트레인은 D452-2(MATa , leu2 , his3 , ura3 , canl), L2612(MATa , leu2 -3, leu2 -112, ura3 -52, trpl -298, canl , cynl , gal +) 및 CEN.PK 이다. 자일리톨, 아세테이트 및 에탄올의 생산은 자일로스 및 OD600 의 사용과 함께 모니터하였다. 결과는 D452-2 스트레인이 세 가지 실험한 스트레인 중에서 최고였다(도 42-44). S. 세레비시아 D452-2 는 효모에서 자일로스-대사작용하는 효소의 유전자 조작에 사용되었다. 본 연구에서 사용된 스트레인 및 플라즈마를 표 17 에 나타내었다.
[표 17]
유전자 조작된 자일로스-발효된 S. 세레비시아 스트레인(DA24)는 자일로스를 소비하고 무시해도 될 정도의 양(negligible amounts)의 자일리톨 축척을 하면서 에탄올을 생산하였다. 40 및 80 g/L 의 자일로스를 단일 탄소원으로서 사용하였을 때, DA24 스트레인은 둘 다 진탕-플라스크 및 바이오리액터 발효 실험에서 일관된 수득률(consistent yields)(Y에탄올/ 자일로스 = 0.31- 0.32 g/g)을 갖는 에탄올을 생산하였다(도 45). 그러나, DA24 스트레인은 자연 발생적으로 존재하는 자일로스-발효 효소, P. 스티피티스 보다 자일로스 소비가 보다 더 늦어졌다. DA24 의 자일로스 발효 능력(Xylose fermentation capability)은 진화하는 유전자 조작된 접근법(evolutionary engineering approach)을 사용하여 추가적으로 개선되었다(Sauer 2001). 자일로스-포함된 배지에서 DA24 의 반복된 계대-배양(sub-cultures) 후에 분리된 하나의 스트레인(DA24-16)을, 다양한 배양 조건 하에서 부모 스트레인(parental strain)과 비교하였을 때, 보다 더 빠른 자일로스 발효 비율을 나타내었다(표 18).
[표 18]
흥미롭게도, DA24-16 스트레인은 P. 스티피티스 , 공지된 가장 빠른 자일로스-발효 효모 만큼 빨리 자일로스를 소비하였다. 그러나, DA24-16 에 의한 에탄올 수득률은 P. 스티피티스 보다 약간 더 낮았다(도 46).
스크린(screen)을, 게놈 라이브러리(genomic library)로 형질전환된(transformed) 자일로스-활용 효소(스트레인 YSX3)가 발현된 S. 세레비시아 스트레인 L2612 를 사용하여 준비하였다(set up). 형질전환한 다음에, 자일로스 사용에 효율적인 스트레인에 대해서 풍부하게 하기 위해서 산소-제한 조건 하에서 40 g/L 자일로스에서 시리얼 배양 이전(serial culture transfer) 하였다. OD600 = 10 에 도달하였을 때, 발효를 산소-제한 조건 하에서 50 mL 의 YPX 배지에서 실행하였고, 0.1 %(50 μL)의 완전히 성장한 세포 배양물을 그 다음 시리얼 배양물(the next serial culture)에 이동시켰다. 10 번의 시리얼 배양한 후에, 세포를 YPX(40 g/L) 한천 배지에서 단계 희석법(serial dilution)으로 확산시켰다(spread). 5 mL 의 YPX 배지를 사용한 발효 실험을 통해서, 낮은 자일리톨 및 높은 에탄올 생산을 하는 콜로니를 스크린하였다(screened). DNA 시퀀싱(DNA sequencing)은 두 가지의 가장 효율적인 스트레인은 XYL2 의 통합된 복사본(integrated copies)을 포함하고, 이는 상동 재조합(homologous recombination)을 통해서 다중-복제 플라스미드(multi-copy plasmid) 내로 클론되었고(cloned), YSX3 세포 내로 형질전환되었다(transformed).
XYL2 유전자는 상이한 강도의 프로모터 조절 하에서 통합 벡터, 예를 들어 TDHp 또는 PGKp 에 두었고, YSX3 세포 내로 형질전환시켰다(도 47). 형질전환된(transformed) 효모 세포에서 자일리톨 및 에탄올 생산에 대한 이러한 플라스미드의 효과를 모니터하기 위해, 연구를 실시하였다. XYL2(PGKp 하에서)의 보다 높은 수준이 발현된 YSX3 세포는 에탄올 생산에 보다 효율적이었고, 게다가 보다 낮은 양의 자일리톨이 생산된 결과를 나타내었다(도 48). 추가적인 XYL3 은 이러한 세포(SRI 스트레인으로 명명)에서 발현되었을 때, 생산된 자일리톨의 양은 결과적으로 스트레인 SRu-23 에서 추가적으로 감소되었다(도 49). 따라서, 유전자 조작된 S. 세레비시아 스트레인에서 XYL2 발현 수준은 자일로스 발효를 시행하기 위한 중요한 요인(key factor)이고, 강한 프로모터 하에서 발현하였을 때, 스트레인은 높은 에탄올 수득할 뿐만 아니라 보다 낮은 자일리톨 축적을 갖는다. XYL2 및 XYL3 의 동시의 과잉-발현은 자일리톨 축적의 양을 추가적으로 감소시킬 수 있다. 그러나 XYL1 은 스트레인 과잉-발현된 XYL2 및 XYL3 에서 추가적으로 과잉-발현되었을 때, 상당한 자일리톨이 축적되었고, 따라서 자일로스 발효가 감소되었다(도 50-51). 따라서, 효율적인 자일로스 발효를 위한 최적 수준의 XYL1 임을 나타내었다.
XYL2 및 XYL3 이 발현된 S. 세레비시아에서 내인성 GRE3 의 과잉-발현이 자일로스 발효를 촉진할 수 있는지 여부를 테스트 하기 위해 실험을 또한 실시하였다. pRS403-GRE3 의 구성에 대해서, GRE3 유전자를 S. 세레비시아 D452-2 로부터 증폭시켰고, TDH3 프로모터 및 CYC 종결자를 갖는 pR403 벡터 내로 삽입하였다. pRS403-GRE3 의 리니얼라이제이션(linearization) 후에, D452-2 의 게놈 내로 이를 통합시켰다. 자일로스-활용 유전자를 효모 스트레인 D452-2 내로 도입하였고(도 52), 자일로스 발효 파라미터(parameters)를 모니터하였다. 특히, 세포가, 높은 OD 접종(inoculations)에서 80g/L 의 자일로스에서 성장하였을 때, GRE3 의 과잉-발현은 에탄올 생산 및 자일리톨 축적에서 XYL1 의 과잉-발현만큼의 효과가 있는 결과를 나타내었다(도 53-54).
유전자 조작된(
engineering
)
LAD
및
XDH
NADPH-의존적인 XR 및 NAD+-의존적인 XDH 및 LAD 사이의 보조인자 불균형에 의해 기인하는, L-아라비니톨 및 자일리톨 축적은 오탄당-이용(pentose-utilizing) 효소가 발현된 유전자 조작된 S. 세레비시아에서의 자일로스 발효 동안에 주요한 장애물(major bottleneck)로서 간주된다. XR 과 XDH 사이의 불균형은 역전된 보조인자 선호(reversed cofactor preferences)를 갖는 유전자 조작된 효소에 의해 정정되는 반면에(Watanabe 등의, 2007; Matsushika 등의, 2008; Bengtsson 등의, 2009), 수정된 효소(modified enzymes)가 감소된 특이적인 활성도를 갖는 바와 같이, 이러한 접근은 감소된 플럭스(reduced flux)를 야기한다. NADH 상에서 NADPH 에 대한 보다 낮은 선호를 나타낸 반면에, 야생형 psXR 은 NADPH 에 대한 2-배(two-fold) 보다 높은 선호를 나타냄이 P. 스티피티스 XR 돌연변이체에서 보고되고 있다(Watanabe 등의, 2007).
이러한 연구에서, 보조인자 특이성을 변경하기 위해 N. 크라사로부터 L-아라비니톨 4-탈수소효소(LAD) 및 XDH 에서 유사한 연구를 실시하였고, 이런 이유로 유전자 조작된 S. 세레비시아에서 자일로스 발효가 개선되었다.
추정상의
LAD
-코딩 유전자의 확인
추정상의 LAD-코딩 유전자의 확인 방법 및 LAD-코딩 유전자 및 추정상의 LAD-코딩 유전자를 클로닝하는 방법을 기재하였다.
추정상의
LAD
-코딩 유전자의 확인
프로브로서 ncLAD(EAA36547.1)을 사용한 단백질 BLAST 검색으로부터, 두 가지의 추정상의 유전자를 각각 P. 크리소게늄 (P. chrysogenum )(XP_002569286.1) 및 P. 귈리어몬디( P. guilliermondii )(EDK37120.2)에서 확인하였다. ncLAD 을 갖는 이러한 두 가지 단백질의 아미노산 동일성은 각각 71 % 및 46 % 이었다.
클로닝
LAD
-코딩 및
추정상의
LAD
-코딩 유전자
A. 니가(NRRL 326), P. 귈리어몬디(NRRL Y2075) 및 P. 크리소게늄(NRRL 807)를 United States Department of Agriculture Agricultural Research Service Culture Collection(미국 일리노이주 피오리아에 위치)로부터 입수하였다. T. 롱 기브라키 아텀(T. longibrachiatum )(T. 레세이 , YSM 768)은 German Resource Centre for Biological Material 로부터 입수하였다(DSMZ).
A. 니가 , T. 롱기브라키아텀 , P. 크리소게늄 및 P. 귈리어몬디를 1 % 효모 추출물, 2 % 펩톤 및 2 % L-아라비노스를 포함하는 한천 플레이트(agar plates) 또는 액체 배지에서 성장시켰다. 세포를 전체 RNA 또는 게놈 DNA 의 분리를 위해 액체 질소에서 세포를 냉동시켰다. cDNA 를 수득하기 위해, 역 전사-PCR(RT-PCR)을 T. 롱기브라키아텀 , P. 크리소게늄 및 P. 귈리어몬디로 부터 분리된 mRNA 에서 실행하였고, PCR 을 (추정상의) LADs 를 코딩하는 유전자를 수득하기 위해 사용하였다. A. 니가에 대해서, 추정상의 LAD 유전자를 알려지지 않은 이유 때문에 cDNA 로부터 증폭시키지 않았다. 따라서, 중복 확장-PCR(overlap extension-PCR, OE-PCR)은 분리된 게놈 DNA 로부터 이러한 인트론-포함된 유전자를 클론하는데 사용하였다. 참고로 이러한 유전자는 클론하는데 사용된 모든 프라이머 서열을 표 19 에 작성하였다.
[표 19]
PCR 산물을 pET-28a 벡터 내로 서브클론하였고(subcloned), 구조물(constructs)을, 각각 클로닝 및 발현을 위한 전기천공법(electroporation)에 의해서 두 가지의 E. coli 스트레인, DH5a 및 BL21(DE3) 내로 형질전환시키는데 사용하였다. NdeI/BamHl 제한 부위(restriction sites)를 A. 니가 , T. 롱기브라키아텀 및 P. 귈리어몬디로부터의 예측된 유전자의 서브클로닝(subcloning)에 대해서 사용하였고, Ndel/ EcoRI 사이트는 P. 크리소게늄에 대해서 사용하였다. 구조물(constructs)을 N-말단 His6-태그된(tagged) 융합물(fusions)로서의 (추정상의) LADs 를 코드하였다. 플라스미드를, 일리노이 대학 어배너-샴페인에서의 Biotechnology Center 에서의 BIGDYE(TM) 종결자 시퀀싱 방법(Terminator sequencing method)을 사용하여 배열 순서를 밝히고(sequenced), 3730xL Genetic Analyzer(미국 캘리포니아주 포스터 시티에 위치한 Applied Biosystems)로 분석하였다.
단백질 발현 및 정제
pcLAD(XP_002569286.1), pgLAD(EDK37120.2), anLAD(CAH69383.1) 및 tlLAD(AAL08944.1)을 코딩하는 유전자를 pET-28a 벡터 내로 클론하였고, E. coli BL21(DE3)에 발현시켰다. LAD 유전자를 포함하는 E. coli BL21(DE3)를, 250 rpm 으로 회전식 진탕기(rotary shaker)에서 30 ℃ 에서 밤새 성장시켰다. 밤새 배양물(50 μL)를 신선한 배양물(5 mL)을 배양하는데 사용하였고, 이를 600 nm(OD600)에 최적의 밀도가 0.6-1.0 에 도달할 때까지, 30 ℃ 에서 250 rpm 으로 교반시켜 성장시켰다. 그리고 난 다음에 배양물을 3-4 시간 동안 30 ℃ 로 또는 20 시간 동안 18 ℃ 로 0.3 mM IPTG 로 배양시켰다.
유도된 세포(1 mL)를, 1 mg/mL 의 라이소자임(lysozyme)을 갖는 1 mL 의 50 mM 인산칼륨 완충용액(pH 7.0)에 이들을 재-현탁시키고, 30 ℃ 에서 30 분 동안 250 rpm 으로 교반시키면서 용해시켰다(lysed). 세포를 밤새 - 80 ℃ 에서 유지시키고, 실온에서 용해시켰다(thawed). 결과적으로 생성된 세포 분해물(cell lysates)을 15 분 동안 13,200 rpm 으로 원심분리하고, 상청액 및 침전물을 도데실 황산 나트륨-폴리아크릴아미드 겔 전기영동(sodium dodecyl sulfate-polyacrylamide gel electrophoresis, SDS-PAGE)에 의한 단백질 발현에 대해서 분석하였다.
단백질 정제에 대해서, 유도된 세포(induced cells)(400 mL)를 1 mg/mL 의 라이소자임(lysozyme)을 갖는 15 mL 의 완충용액 A (20 mM Tris, 0.5 M NaCl, 20% 글리세롤, pH 7.6)로 처리하고, 30 ℃ 에서 30 분 250 rpm 으로 교반시켰다. 냉동-융해 사이클(freeze-thaw cycle) 후에, 결과적으로 생성된 생산물을 추가적으로 음파처리(sonication)로 용해시킨 후에, 세포 잔유물(cell debris)을 제거하기 위해 20 분 동안 12,000 rpm 으로 원심분리하였다. 제조자의 지시에 따라서 His6-태그된 단백질을 정제하기 위해 Co2 +-고정화(immobilized) 금속 친화도 크로마토그래피 수지로 가득찬 컬럼에 상청액을 적용시켰다. 정제된 단백질을 초미세여과(ultrafiltration)(미국 매사추세츠주 빌러리카에 위치한 Millipore, Amicon Ultra)으로 탈염하였고(desalted), 150 mM NaCl 및 15 % 의 글리세롤을 포함하는 HEPES 완충용액(pH 7.0)으로 세척하였고, - 20 ℃ 에서 유지시켰다. 단백질 농도를 제조자의 프로토콜에 따라서 브래드퍼드법(Bradford method)(Bradford 1976)에 따라 결정되었다.
LAD
단백질의 특성
LAD 효소의 정상-상태 반응속도(steady-state kinetics), 분자량, 4 차 구조(quaternary structure), 온도 의존성, pH 의존성, L-아라비니톨 탈수소효소 활성도 및 금속 함량을 분석하였다.
L-
아라비니톨
탈수소효소 활성도
P. 크리소게늄 , P. 귈리어몬디 , A. 니가 및 T. 롱기브라키아텀으로부터의 LAD 발현된 숙주 세포로부터 용해물(Lysate)을 제조하였다. 10 마이크로리터의 세포 용해물(cell lysate)을 50 mM 의 인산칼륨 완충용액(pH 7.0)에서의 기질로서 200 mM L-아라비니톨 및 2 mM NAD+ 과 함께 활성도 검정에 사용하였다. NADH 생산을 Cary 300 Bio UV-vis 분광 광도계(미국 노스캐롤라이나주 캐리에 위치한 Varian)를 사용하여 340 nm(ε = 6.22 mM-1cm-1)에서 흡광도를 측정하여 모니터하였다.
정상-상태 반응속도(
Steady
-
State
Kinetics
)
상이한 LAD 효소의 동역학적 파라미터를 측정하였다. 초기의 비율을 50 mM 인산칼륨 완충용액(pH 7.0)에 실온에서 UV-vis 분광광도계(spectrophotomer)를 사용한 340 nm 에서 흡광도 변화(absorbance change)를 측정하여 결정하였다. 초기의 비율을, 다양한 농도의 기질(L-아라비니톨) 및 보조인자(NAD+/NADP+)(L-아라비니톨에 대해서 5 내지 320 mM, 보조인자에 대해서 0.5 내지 3.2 mM)에서 측정하였다. 기질 및 보조인자의 효소 반응속도를 미하엘리스-멘텐 반응속도론(Michaelis-Menten kinetics)을 사용하여 분석하였고, 동력학적 파라미터(kinetic parameters)를 라인웨버-버크도면(Lineweaver-Burk plot)에 데이터를 맞추어 결정하였다. 기질에 대한 파라미터를 포화 보조인자 농도(3.2 mM)에서 초기의 비율을 측정하여 결정하였고, 보조인자에 대한 이러한 비율을 포화 기질 농도(320 mM)에서 결정하였다. 검정을 3 번 실시하였다.
클론된 LADs 를 L-아라비니톨에 대해서 상이한 결합 친화도(binding affinities) 및 촉매 활성도(catalytic activities)를 나타내었다 : K m 은 2 배 달랐고, K cat 는 LADs 중에서 약 3 배나 달랐다. L-아라비니톨에 대해서, anLAD, tlLAD, 및 pcLAD 의 K m 값은 각각 25 ± 1, 18 ± 1 및 37 ± 2 mM 이고, k cat 값은 각각 507 ± 22, 346 ± 41 및 1085 ± 71 min-1 이다(표 20). tlLAD 효소가 가장 낮은 K m 을 가진 반면에, pcLAD 는 가장 높은 K m 값을 가짐에도 불구하고, 가장 높은 촉매 활성도(k cat ) 및 효율성( k cat / K m )을 나타내었다(표 20). 보조인자 NAD+ 속도에 대해서, 클론된 LADs 는 0.2-0.3 mM 의 범위에서 K m 값 및 2526 내지 3460 mM-1?min- 1 의 범위에서 촉매 효율성(catalytic efficiencies)을 나타내었다(표 21). 모든 클론된 LADs 는 NADP+ 에 대해서 최소한의 활성도를 나타내었다(표 20, 21). 초기의 비율은, 광범위한(large) K m 때문에, 가장 높은 기질 및 보조인자 농도(L-아라비니톨에 대해서 320 mM 및 NADP+ 에 대해서 3.2 mM)에서 포화되지 않았다. 따라서, 효소의 촉매 효율성만이 NADP+ 에 대해서 0.1 또는 0.2 mM 및 L-아라비니톨에 대해서 10 또는 20 mM (K m >> [S])으로 결정되었다(표 20, 21).
[표 20]
[표 21]
분자량 및 4 차 구조
4 가지의 단백질의 서브유닛(subunits)의 계산된 분자량은 43 kDa (anLAD), 41 kDa (tlLAD), 42 kDa (pcLAD), 및 42 kDa (pgLAD)이다. 단백질의 분자량을 Shimadzu HPLC 시스템(일본 교토에 위치한 Shimadzu)에서 Bio-Sil SEC-250 컬럼(300x7.8 mm, 미국 캘리포니아 허큘러스에 위치한 Bio-Rad)을 사용하여 결정하였다. 이동상(mobile phase)은 50 mM Na2HP04, 50 mM NaH2P04, 150 mM NaCl 및 10 mM NaN3(pH 6.8)으로 구성되어 있고, 유량(flow rate)은 1.0 mL/min 이다. 분자량을 표준 단백질 분자량의 보존시간(retention times)과 비교하여 측정하였다.
4 차 구조를 HPLC 및 단량체 서브유닛의 분자량에 의해 관찰된 분자량을 기반으로 측정하였고, 이를 SDS-PAGE 분석으로 결정하였다. an-, tl-, 및 pcLAD 의 분자량은 각각 178, 194 및 173 kDa 으로 측정되었다. SDS-PAGE 로 결정된 서브유닛(subunits)의 분자량을 비교하여, LADs 는 이들의 천연형태(native forms)에서 비-공유결합으로 연결된 사합체(tetramers)이다.
온도 및
pH
의존성
단백질의 최적의 온도를 10 내지 70 ℃ 범위의 온도에서 효소 활성도를 검정하여 결정하였다. 열 불활성을, 인산 완충용액에서 50 ℃ 에서 다양한 배양 시간 후에 효소 활성도(enzyme activity)를 측정하여 결정하였다. 효소 활성도를 2 mM NAD+ 및 200 mM L-아라비니톨로 측정하였다. 효소 활성도의 반감기(Half-life)를 첫 번째-오더 지수 붕괴 함수(first-order exponential decay function)를 사용하여 측정하였다. 온도를 UV-vis 분광 광도계에 연결된 Cary 온도 컨트롤로(Cary temperature controller)(미국 노스캐롤라이나주 캐리에 위치한 Varian)에 의해 조절하였다. pH-의존적 효소 활성도는, 일반적인 완충용액(50 mM 모르폴린에탄술폰산/50 mM 트리스/50 mM 글리신)에서 NAD+ (2 mM) 및 L-아라비니톨(200 mM)의 포화 농도에서 5.0 과 11.0 사이의 pH 에서의 활성도를 측정하여 결정하였다(Ellis and Morrison 1982).
anLAD 및 pcLAD 의 최적 온도는 40 에서 50 ℃ 사이인 반면에, tlLAD 은 55 에서 65 ℃ 사이의 보다 높은 최적 온도를 나타내었다(도 55A). LADs 의 촉매 활성도는 50 ℃ 에서 배양 시간의 길이(length)에 따라 기하급수적으로 감소되었고, 100 분 후에 거의 대부분 완전하게 불활성화되었다(deactivated)(도 55B). tlLAD 는 50 ℃ 에서 20 분의 반감기를 가지면서 열에 의해 가장 안정되었고, anLAD 는 50 ℃ 에서 5 분 미만의 반감기를 가지면서 가장 낮게 안정되었다. 모든 특성화된 LADs 는 약 pH 9.4 에서 최대의 활성도를 갖는 pH 범위 7 내지 11 에서 활성(activity)을 나타내었고(도 55C). pH 범위 9 내지 10 외에서, 활성도는 현저하게 감소되었고, 대략 20 % 의 활성도가 pH 7.0 에서 남아있었다(도 55C). pH 5.0 이하에서는 어떠한 활성도도 발견되지 않았다.
금속 분석(
Metal
analysis
)
금속 분석에 대한 사본 샘플(Duplicate samples)을 완충용액 교환 및 동결건조(lyophilization)에 의한 인산완충식염수(phosphate buffered saline, PBS)에서 제조하였다. 각각의 샘플은 1 mL 의 완충용액에 1-2 mg 의 단백질을 포함한다. 금속의 함량 및 정체(identity)를 Microanalytical Laboratory at the University of Illinois at Urbana-Champaign(미국 일리노이드주 어배나에 위치)에서의 고주파유도결합형 플라즈마 발광 분석법(inductively coupled plasma atomic emission spectrometry)(OES Optima 2000 DV, 미국 매사추세츠주 보스턴에 위치한 Perkin Elmer)으로 분석하였다.
Zn2 + 의 분자량 퍼센트는 1 : 1 분자비(molar ratio)를 기반으로 계산한 것에 가까웠다(표 22).
[표 22]
변경된 보조인자 특이성(
Altered
Cofactor
Specificity
)을 갖는
LAD
효소의 유전자 조작(
engineering
)
LADs 의 변경된 보조인자 특이성의 방법을 결정하였고, 돌연변이된 LADs 는 변경된 보조인자 특이성 및 그 밖의 특성에 대해서 분석하였다.
변경된 보조인자 특이성을 갖는
LADs 의
개발(
Development
)
NAD+ 에서 NADP+ 까지의 anLAD, tlLAD 및 pcLAD 의 보조인자 특이성을 변경하기 위해 부위 특이적 돌연변이(Site-directed mutagenesis)를 실시하였다. 변경된 보조인자 특이성을 갖는 tlLAD 를 생성하기 위해, 자연적으로 발생한 tlLAD 의 아미노산 번호 224, 225 및 362 을 각각 세린, 아르기닌 및 트레오닌으로 치환하였다. 클론된 anLAD 및 pcLAD 의 아미노산 서열은 T. 롱기브라키아텀LAD (tlLAD) 서열과 정렬하였고(aligned), tlLAD 아미노산 번호 224, 225 및 362 에 부합하는 아미노산이 돌연변이 되었다(mutated). 변경된 보조인자 특이성을 갖는 모든 LADs 에 대해서, 조효소 결합 도메인의 β-α-β 모티프(motif) 내의 두 개의 아미노산 잔기가 세린 및 아르기닌으로 각각 교체되었고 ; anLAD 에 대해서 D213 및 1214, tlLAD 에 대해서 D224 및 1225, 및 pcLAD 에 대해서 D212 및 1213(Korkhin 등의, 1998; Pauly 등의, 2003; Watanabe 등의, 2005), 세 번째 돌연변이를 anLAD 에 대해서 A359, tlLAD 에 대해서 A362 및 pcLAD 에 대해서 S358 에 도입되었고, 트레오닌으로 교체되었다(프라이머 서열에 대해서, 표 23 참조). 메가프라이머(Megaprimer) PCR 방법을 주형(templates)으로서 야생형 LAD 구조물(constructs)을 사용한 부위-특이적인 돌연변이(site-specific mutations)를 도입하여 사용하였다(Sarkar and Sommer 1990). 정확한 돌연변이를 DNA 서열 분석(DNA sequence analysis)으로 확인하였다.
[표 23]
변경된 보조인자 특이성을 갖는
LADs 의
동력학적 분석(
Kinetic
Analysis
)
본원의 실시예에서, "tlLAD 돌연변이체(mutant)"는 돌연변이 D224S / I225R / A362T 를 갖는 tlLAD 로서 규정하였고 ; "anLAD 돌연변이체"는 돌연변이 D213S / I214R / A359T 를 갖는 anLAD 로서 규정하였고 ; "pcLAD 돌연변이체"는 돌연변이 D212S / I213R / S358T 를 갖는 pcLAD 로서 규정하였다. tlLAD 돌연변이체는 NAD+ 에서 NADP+까지의 현저하게 변경된 보조인자 특이성을 나타내었다. 이는 가장 높은 촉매 활성도를 또한 나타내었다. NADP+ 를 갖는 L-아라비니톨에 대한 tlLAD 돌연변이체의 K m 및 K cat 는 각각 46 ± 4 mM 및 170 ± 9 min-1 이다(표 24). 포화된 NAD+ 를 갖는 tlLAD 돌연변이체를 포함하는 모든 검정에서, 반응 속도의 정체기(plateau)는 실험된 농도 범위에서는 관찰되지 않았고, 따라서 촉매 효율성(catalytic efficiencies)은 NAD+ 에 대해서 0.8 mM 및 L-아라비니톨에 대해서 80 mM 으로 측정되었다(표 24, 25). 보조인자에 대해서, anLAD 및 tlLAD 돌연변이체는 NAD+ 상에서 NADP+ 에 대해 현저하게 보다 높은 선호도를 나타내었다(표 25). anLAD 및 tlLAD 돌연변이체의 K m 값은 각각 0.46 ± 0.09 및 0.10 ± 0.01 mM 이고, K cat 값은 각각 55.7 ± 6.4 및 90.5 ± 9.2 min-1 이다(표 25). anLAD 및 tlLAD 돌연변이체의 촉매 효율성(catalytic efficiencies)은 130 ± 32 및 934 ± 72 mM-1?min-1 이고, NAD+ 에 대한 NADP+ 을 갖는 촉해 효율성의 비율은 각각 100 및 161 이다. tlLAD 돌연변이체에 대해서, NAD+ 에 대한 NADP+ 을 갖는 촉해 효율성의 비율은 2.5 x 104 배 증가되었다(표 21, 25). pcLAD 돌연변이체는 NAD+ 와 함께 어떠한 활성도를 나타내지 않았다.
[표 24]
[표 25]
변경된 보조인자 특이성을 갖는 N.
크라사
XDH
(
ncXDH
)의 유전자 조작(
Engineering
)
추정상의
(
putative
)
ncXDH 의
클로닝
(
Cloning
) 및 특성
추정상의 N. 크라사 자일리톨 탈수소효소(ncXDH) 서열을, 의문 서열(query sequence)로서 P. 스티피티스 자일리톨 탈수소효소(psXDH)를 사용하여 National Center for Biotechnology Information 웹사이트(웹사이트 ncbi.nlm.nih.gov)에서 단백질 BLAST 검색을 사용하여 발견하였다. 두 가지의 효소를 ClustalW 알고리즘(algorithm)을 사용하여 완전히 정렬하엿고, 44 % 의 동일성(fully) 및 60 % 의 유사성(similarity)을 공유하는 것으로 나타났다(도 56). 네우로스포라 크라사의 전체-게놈 서열은 발표되었고(Galagan 등의., 2003), 이는 추정상의 자일리톨 탈수소효소(XDH) 유전자의 클로닝에 대한 프라이머를 디자인하는데(design) 사용되었다.
D-자일로스-유도된 N. 크라사 10333 으로부터 분리된 전체 RNA 에서 실행한 RT-PCR 은 예측된 크기의 유전자 산물(-1.1 kb)을 나타내었다. RT-PCR 산물은 Ndel 및 Sacl 제한효소 부위(restriction sites)를 사용한 pET-28a 벡터 내로 클론되었고, E. coli BL21 (DE3) 내로 형질전환되었다(transformed). 이러한 구조물(construct)(pET-28a ncXDH)은 트롬빈 분해 사이트(thrombin cleavage site)를 갖는 N-말단 His6-태그된 융합물(fusion)로서의 ncXDH 가 발현되었다. 이러한 세포의 IPTG-유도된 배양물의 세포 용해물(Cell lysates)을 제조하였고, SDS-PAGE 로 분석하였고, XDH 활성도에 대해서 검정하였다. 제조자의 프로토콜에 따라서, Talon® Co2+ 슈퍼플로우 수지(Superflow resin)(Clontech, Mountain View, CA)를 사용한 고정화 금속 이온 친화도 크로마토그래피(immobilized metal ion affinity chromatography, IMAC)로 XDH 를 정제하였다. 정제된 단백질을 50 mM 4-(2-히드록시에틸)피페라진-l-에탄술폰산(HEPES) 완충용액(pH 7.25) + 15 % 글리세롤을 여러 번 세척하면서 초미세여과(ultrafiltration)하여 탈염시키고(desalted), - 80 ℃ 에서 냉동시켜 저장하였다. 단백질 농도를 브래드퍼드법(Bradford method)으로 결정하였다(Bradford 1976).
ncXDH 는 절대적으로 NAD+-선호하는 효소이다. ncXDH 는 높은 안전성(50 ℃ 에서 ~200 분의 반감기) 및 발현을 또한 나타내었다. Watanabe 등의(2005b)에 의한 이전의 작업은 psXDH 의 보조인자 특이성을 역전시키는 것을 목표로한다.
변경된 보조인자 특이성을 갖는
ncXDH 의
개발(
development
)
서열 정렬을 통해서, ncXDH 의 잔기 D204,1205 및 V206 은, ncXDH-ARS 를 생성하기 위해 각각 알라닌, 아르기닌 및 세린에 대한 부위 특이적인 돌연변이를 목표로 한다. 표 26 은, 현재 선호하는 NADP+, ncXDH-ARS 가 완전하게 역전된 보조인자 특이성을 갖음을 나타낸다. 기질 자일리톨에 대한 친화도(affinity)는 보조-인자에 대한 친화도-변화로부터 매우 많이 악화되지 않았다.
[표 26]
ncXDH
돌연변이체의 동역학적 분석(
kinetic
analysis
)
돌연변이체 ncXDH 는 보조인자 특이성의 극적인 전환(dramatic reversal)을 갖는다. NADP+ 에 대한 돌연변이체 ncXDH 의 K m 은, NAD+ 에 대한 야생형 ncXDH 의 K m 보다 약 2.5-배 보다 높은 반면에, K cat 값은 유사하였다(표 27).
[표 27]
도 57 에 나타낸 바와 같이, XDH 활성도는 pH 4.0 에 이르기까지 확장된 활성도를 갖는 보다 산성인 조건에 대한 보다 높은 내성(tolerance)을 나타낸 반면에, LAD 활성도는 생체외에서 활성도 검정에서 pH 5.0 에서 정지하였다(abolished).
S.
세레비시아에서
박테로이드
스테로코리스(
Bacteroides
stercoris
)로부터의 자일로스
이성질화효소(xylose isomerase)의
발현
박테리아 자일로스 이성질화효소(XI)는 자일로스에서 자일루로스의 전환을 포함한다. 최근에는, 혐기성 곰팡이의 두 가지 종[피로마이세스종 ( Piromyces sp .) 및 오르피노마이세스종( Orpinomyces sp .)] 및 혐기성 박테리아[클로스트리튬 파이토퍼먼탄스( Clostridium phytofermentans )]로부터의 활성형 XI 가 발현된 세 가지의 성공적인 경우가 보고되고 있다. 피로마이세스 종 E2 으로부터의 균류에 의한 XYLA 유전자는 S. 세레비시아에서 기능적으로 발현되었고, XI 활성도의 최대한의 1.1 U/mg-단백질을 30 ℃ 에서 수득하였다(Kuyper 등의, 2003). 피로마이세스 종으로부터의 유전자와 94 % 동일성을 갖는, 오르피노마이세스로부터의 두 번째 균류에 의한 XYLA 유전자는 S. 세레비시아에서 또한 기능적으로 발현되었다(Madhavan 등의, 2009). 최근에는, 클로스트리튬 파이토퍼먼탄스로부터의 첫 번째 원핵세포의 xylA 유전자가 S. 세레비시아에서 기능적으로 발현되었다(Brat 등의., 2009).
혐기성 박테리아 박테로이드 스테로코리스(BtXI)로부터의 이성질화효소(isomerase) xylA 는 피로마이세스 종으로부터의 이성질화효소와 높은 서열 동일성을 공유한다(82 %). BtXI 를 pRS424TEF 벡터 내로 클론시켰고, S. 세레비시아 L2612 스트레인 내로 형질전환시켰다(transformed). 유전자는 pRS403TEF 벡터를 사용하여 S. 세레비시아 D452-2 내로 또한 통합되었다. 에탄올 생산을 BtXI (L2612 에서 5g/L 및 D452-2 에서 7.8 g/L)가 발현된 둘 다의 스트레인에서 관찰되었다(도 58-59). 그러나, 생산의 비율은 XYL 유전자가 발현된 유전자조작된 스트레인의 비율과 비교하여 상대적으로 낮았다.
낮은 에탄올 생산은 어떠한 축적된 자일리톨(내인성 효소 알도스 환원효소에 의해 자일로스로부터 형성)의 억제적 효과에 기인할 수 있다. 자일리톨 축적을 감소시키기 위해서, XDH 및 XK 는 BTXI-발현된 효모 스트레인에서 발현되었다(DBtXI). 결과적으로 생성된 스트레인은 약간의 개선된 에탄올 수득을 갖고, 감소된 자일리톨 생산을 갖는다(도 60). DBtXI 에서 이러한 두 가지 XYL 유전자의 공동-발현(Co-expression)은 혐기성 조건 하에서도 에탄올 생산을 야기한다.
오탄당 인산 경로에서의 효소의 과잉-발현(
Over
-
Expression
)
PPP 효소 P. 스티피티스로부터의 글루코스-6-포스페이트 탈수소효소(ZWF1), 6-포스포글루코네이트 탈수소효소(GDN1), 트랜스알돌라아제(TAL1) 및 트랜스케톨라아제(TKT1)는 강한 프로모터(strong promoter)(PGPD)의 조절 하에서 통합 벡터(pRS406) 내로 클론되었다(cloned). 플라스미드를 효소 StuI 에 의해 선모양으로 만들고(linearized), S. 세레비시아의 염색체(chromosome) 내로 통합되었다.
그러나, 과잉-발현된 PPP 효소의 유익한 효과를 얻기 위해서, XYL3(XK)의 과잉-발현될 수 있는 것을 또한 갖는다(도 61). XYL3 및 PPP 효소의 발현은 YP-자일루로스 배지에서 에탄올 생산을 또한 증진시킨다.
알도스
-1-
에피머화효소(Epimerase)의
발현
β-글루코스다아제에 의한 셀로비오스의 가수분해(Hydrolysis)는 β-D-글루코스를 방출하였다. 그러나 효모 헥소키나아제(hexokinases)는 α-D-글루코스를 선호하고(또는 배타적인 사용), β-D-글루코스에서 α-D-글루코스로의 무타로에이션(mutaroation)의 비율은 대사율(metabolic rate)을 효율적으로 늦출 수 있다. 이러한 전환(conversion)을 증진시키는 하나의 방법은 예측된 알도스-1-에피머화 효소 NCU09705 를 과잉-발현시킬 수 있다. 이러한 가설(hypothesis)은 과잉-발현된 NCU09705 상동성(homologs)에 의해 테스트되었다 : S. 세레비시아에서의 GAL10, YHR210C 및 YNR071C ; 및 P. 스티피티스에서의 GAL 10. 스트레인을 셀로비오스 소비 및 에탄올 생산에 대해서 테스트하였다(도 62). 결과는, S. 세레비시아에서 상동성의 과잉-발현(over-expression)은 셀로비오스 소비 및 에탄올 생산에서 약간의 증가됨을 결과는 나타내었다.
자일로스
및
셀로비오스의
공동-발효(
Co
-
Fermentation
)
이러한 실시예에서, 글루코스의 이합체인, 셀로비오스가 자일로스(오탄당)와 함께 공동-발효된 곳에서 글루코스 억제를 극복하기 위한 새로운 전략이 사용되었다. 셀로비오스는 셀룰로스의 효소의 가수분해로부터의 중간 생산물(intermediate product)이고, 이는 엑소셀룰라아제(exocellulases), 엔도셀룰라아제(endocellulases) 및 β-글루코시다아제를 포함하는 셀룰라아제의 칵테일(cocktail)에서 β-글루코시다아제에 의해 글루코스로 추가적으로 전환될 수 있는 반면에, 오탄당 당은 헤미셀룰로스(hemicellulose)의 희석된 산 가수분해(dilute acid hydrolysis)의 산물이다. 셀로비오스를 글루코스로 가수분해할 수 있는β-글루코시다아제 및 셀로비오스 운반체 둘 다가 결핍되어 있기 때문에 야생형 S. 세레비시아는 셀로비오스를 축적할 수 없다. 이런 이유로, 최근에 발견된 셀로덱스트린 운반체 유전자를 실시예 9 에 나타내었고, N. 크라사로부터의 β-글루코시다아제 유전자가 S. 세레비시아에서 공동-발현되었고, 자일로스 및 셀로비오스의 혼합물을 탄소원으로서 사용하였다(도 63). S. 세레비시아로부터의 셀로비오스 발효를 가능하게 하기 위해, 유사한 접근은 β-글루코시다아제의 분비(secretion) 또는 세포 표면 전시(cell surface display)를 사용하였다(van Rooyen 등의, 2005; Skory 등의, 1996; Kotaka 등의, 2008; Katahira 등의, 2006). 이러한 경우에, 셀로비오스는 S. 세레비시아의 내인성 육탄당 수송 시스템(endogenous hexose transport system)에 의해 수송되기 전에 세포 밖에서(extracellularly) 글루코스로 가수분해하였다. 그에 반해서, 이러한 전략에서, 셀로비오스는 세포 내로 수송된 다음에 가수분해되었다.
S. 세레비시아에서 혼합된 당 발효를 위한 일반적인 방법에서, 리그노셀룰로스(lignocellulose)로부터 유도된 글루코스 및 오탄당 당(pentose sugars)의 혼합물을 사용하였다. 그러나, 이러한 새로운 전략(strategy)에서, 셀로비오스 및 오탄당 당의 혼합물을 사용하였다. 셀로비오스는 이종의 셀로덱스트린 운반체(heterologous cellodextrin transporters)를 통해 효모 세포 내로 수송된 반면에, 오탄당 당은 내인성 육탄당 운반체(endogenous hexose transporters)에 의한 효소 세포 내로 수송되었고, 이로 인하여 글루코스 저해에 부분적으로 책임이 있는 페노메논(phenomenon)인, 동일한 운반체에 의한 글루코스 및 오탄당 당 사이의 직접적인 경쟁이 제거되었다. 효모 세포 내에서 일단은, 셀로비오스는 β-글루코시다아제에 의해 글루코스로 전환되고, 효모 세포에 의해 즉시 소비되고, 그 결과로 이는 세포내 글루코스 농도가 낮아지는 결과가 발생하고, 이로 인하여 추가적으로 글루코스 저해가 완화되었다.
유전자 조작된(engineered) 자일로스-이용 효모 스트레인 L2612 은 셀로덱스트린 운반체 및 β-글루코시다아제 유전자가 공동-발현되는(co-express) 숙주(host)로서 사용되었다. 이러한 스트레인에서, 자일로스 환원효소 및 자일리톨 탈수소효소 및 피치아 스티피티스로부터의 자일루로키나아제(xylulokinase)로 구성된 D-자일로스 이용 경로(utilization pathway)는 염색체 내에 통합되었다. NCU008011, NCU08114 및 NCU00809 를 포함하는 네우로스포라 크라사로부터의 셀로덱스트린 운반체, 및 네우로스포라 크라사로부터의 하나와 아스페르길루스 아쿨레 아투스( Aspergillus aculeatus)로부터의 다른 하나를 포함하는 두 개의 β-글루코시다아제 유전자를 평가하였다(evaluated).
플라스미드를 유지하기 위해, S. 세레비시아 L2612(MAT α, leu2 -3, leu2-112, ura3 -52, trpl -298, canl , cynl , gal +)를 합성 드롭아웃 배지(synthetic dropout media)에서 배양하였다(아미노산 및 황산암모늄이 없는 0.17 % 의 Difco 효모 질소 염기, 0.5 % 의 황산암모늄 및 0.05 % 의 아미노산 드롭아웃 혼합물). 2 % 당을 함유한 YPA 배지[1 % 의 효모 추출물, 2 % 펩톤, 0.01 % 의 아데닌 헤미술페이트(adenine hemisulfate]는 효모 스트레인을 성장시키는데 사용하였다.
D-자일로스 환원효소, 자일리톨 탈수소효소 및 피치아 스티피티스로부터의 자일루로키나아제(xylulokinase)로 구성된 D-자일로스 이용 경로를 통합하기 위해, 이에 상응하는 유전자를 PCR-증폭하였고, DNA 조립 방법(DNA assembler method)을 사용한 pRS416 플라스미드 내로 클론시켰다(Shao et al ., 2009). BamHl 및 Hindlll 는 D-자일로스 이용 경로를 코딩하는 DNA 단편을 제거하기 위해 사용한 다음에, 동일한 두 가지의 제한 효소(restriction enzymes)에 의해 소화된(digested) pRS406 플라스미드 내로 결합되었다(ligated). 결과적으로 생성된 플라스미드를 Apal 에 의해 선 모양으로 만든 다음에, L2612 의 염색체에서의 URA3 로커스(locus) 내로 통합되었다.
pRS425 플라스미드(미국 매사추세츠주 입스위치에 위치한 New England Biolabs)는 셀로덱스트린 운반체 유전자 및 β-글루코시다아제 유전자가 공동-발현시키는데(co-express) 사용하였다. 도 64 에 나타낸 바와 같이, pRS425 플라스미드를 Bam HI 및 Apal 로 소화시켰다. PYK1 프로모터 및 ADH1 종결자는 각각 셀로덱스트린 운반체의 N-말단 및 C-말단에 첨가한 반면에, TEF1 프로모터 및 PGK1 종결자를 각각 β-글루코시다아제의 N-말단 및 C-말단에 첨가하였다. 이러한 DNA 단편을, DNA 조립 방법을 사용하여 선으로 된(linearized) pRS425 셔틀 벡터 내로 조립하였다(Shao 등의., 2009). 네우로스포라 크라사로부터의 이러한 셀로덱스트린 운반체 유전자 NCU00801(XM_958708), NCU08114(XM_958780) 및 NCU00809(XM_959259), 및 네우로스포라 크라사로부터의 두 개의 β-글루코시다아제 유전자 NCU00130 (XM_951090) 및 아스페르길루스 아쿨레아투스로부터의 BGL1(D64088)를 사용하였다. 하나의 셀로덱스트린 운반체 유전자 및 하나의 β-글루코시다아제 유전자 각각을 갖는 전체로서 6 가지의 조합물(combinations)이 있다.
효모 플라스미드를, 재조합 DNA 조작(recombinant DNA manipulation)에 사용된 E. coli DH5a 내로 이동시켰다(transferred). 형질전환체(transformants)를 00 mg/L 암피실린(ampicillin)을 포함하는 Luria broth 플레이트에 도말하였다(plate). E. coli 형질전환체를, 액체 Luria broth 배지(미국 펜실베이니아주 피츠버그에 위치한 Fisher Scientific) 내에서 배양시킨 다음에 37 ℃ 및 250 rpm 에서 성장시켰다. 플라즈마를 QIAprep Spin Miniprep Kit(QIAGEN)를 사용하여 E. coli 로부터 분리하였다. 이러한 플라스미드를 다음의 스트레인을 수득하기 위해 개별적으로 L2612 스트레인 내에 형질전환시켰다(transformed) : SL01[네우로스포라 크라사로부터의 NCU00801 셀로덱스트린 운반체 유전자 및 NCU00130 β-글루코시다아제 유전자를 포함하는(harboring) 플라스미드를 함유], SL02(네우로스포라 크라사로부터의 NCU00809 셀로덱스트린 운반체 유전자 및 NCU00130 β-글루코시다아제 유전자를 포함하는 플라스미드를 함유), SL03(네우로스포라 크라사로부터의 NCU08114 셀로덱스트린 운반체 유전자 및 NCU00130 β-글루코시다아제 유전자를 포함하는 플라스미드를 함유), SL04(아스페르길루스 아쿨레아투스로부터의 NCU00801 셀로덱스트린 운반체 유전자 및 BGL1 유전자를 포함하는 플라스미드를 함유), SL05(아스페르길루스 아쿨레아투스로부터의 NCU00809 셀로덱스트린 운반체 유전자 및 BGL1 유전자를 포함하는 플라스미드를 함유), 및 SL06(아스페르길루스 아쿨레아투스로부터의 NCU08114 셀로덱스트린 운반체 유전자 및 BGL1 유전자를 포함하는 플라스미드를 함유). SL00 스트레인을 수득하기 위해, 비어있는(empty) pRS425 플라스미드를 L2612 스트레인 내로 형질전환시켰고(transformed), 이를 음성 대조군(negative control)으로서 사용하였다. 효모 형질전환(transformation)을 표준 리튬 아세테이트 방법을 사용하여 실시하였다(Gietz 등의, 1995). 결과적으로 생성된 형질전환 혼합물을 2 % D-글루코스로 보충된 SC-Ura-Leu 배지에 도말하였다(plated).
DNA 조립 방법을 사용한 플라스미드의 적절한 구조를 확인하기 위해, 플라스미드를 Zymoprep Yeast Plasmid Miniprep II 키트(미국 캘리포니아주 오렌지에 위치한 Zymo Research)를 사용하여 효모 세포로부터 분리한 다음에, E. coli DH5a 세포 내로 이동시켰다(transferred). 결과적으로 생성된 세포를 100 mg/L 의 암피실린을 포함하는 LB 플레이트에 확산시켰다(spread). 단일 E. coli 콜로니를 LB 액체 배지 내로 배양시켰다. 플라스미드를 QIAprep Spin Miniprep 키트(미국 캘리포니아주 발렌시아에 위치한 QIAGEN)를 사용하여 E. coli 로부터 분리하였고, 진단(diagnostic) PCR 또는 ClaI 및 HindIII 를 사용한 제한 소화(restriction digestion)로 체크하였다. 모든 제한 효소를 New England Biolabs (미국 매사추세츠주 입스위치에 위치)로부터 입수하였다. 모든 화학물질은 Sigma Aldrich 또는 Fisher Scientific 로부터 입수하였다.
혼합된 당 발효 연구에 대한 충분한 세포를 수득하기 위해, 각각의 효소 스트레인에 대해서, 단일 콜로니를 2 % 글루코스를 더한 2 mL 의 SC-Ura-Leu 배지에서 첫 번째로 성장시킨 다음에, 250 mL 의 진탕 플라스크에 50 mL 의 동일한 배지 내에서 배양시켰다. 하루 성장시킨 후에, 세포를 스핀다운시키고(spun down), 250 mL 의 언배플드 진탕-플라스크(unbaffled shake- flask)에서 4 % 셀로비오스 및 5 % D-자일로스, 또는 4 % 셀로비오스, 5 % 자일로스, 및 0.5 % 글루코스, 또는 4 % 셀로비오스, 5 % 자일로스, 및 1 % 글루코스로 보충된 50 mL 의 YPA 배지에서 배양시켰다. 초기의 OD600 ~1 로부터의 개시하여, 세포 배양물을 산소 제한 조건 하에서 발효를 위해 30 ℃ 에서 100 rpm 으로 성장시켰다. OD600 리딩(reading) 및 세포 배양물 샘플을 다양한 시점에서 채취하였다. 당 농도를 HPLC 를 사용하여 분석한 반면에, 에탄올 형성을 에탄올 키트(독일 다름슈타트에 위치한 R-biopharm)를 사용하여 분석하였다. 각각의 측정점(data point)에 대해서, 세 번의 샘플을 채취하였다. SL00 내지 SL06 까지의 범위의 스트레인에 대해서 혼합된 당 발효 결과를 도 65 에 나타내었다. 최고의 스트레인 SL01 은 추가적인 특성 해석(characterization)을 위해 선별되었다.
SL01 내지 SL066 까지의 6 가지 상이한 스트레인의 전체는, 하나의 셀로덱스트린 운반체 유전자 및 하나의 β-글루코시다아제 유전자를 포함하는 pRS425 플라스미드를 L2612 스트레인 내로 도입시켜서 구성하였다. 각각의 플라스미드에서, 셀로덱스트린 운반체 유전자 및 β-글루코시다아제 유전자는 각각 효모 프로모터 및 종결자에 첨가하였고, DNA 10 조립 방법(DNA 10 assembler method)으로 pRS425 다중-복제 플라스미드(multi-copy plasmid) 내로 조립되었다(Shao 등의, 2009)(도 64). SL00 스트레인을 수득하기 위해 비어있는(empty) pRS425 플라스미드를 L2612 스트레인 내로 도입하였고, 이를 음성대조군으로 사용하였다. 모든 스트레인을 진탕 플라스크에서 40 g/L 셀로비오스 및 50 g/L D-자일로스의 혼합물과 함께 배양하였고, 이들의 당 소비 비율, 세포 성장 비율 및 에탄올 역가(titers)를 결정하였다(도 65). 모든 스트레인 중에서, 네우로스포라 크라사로부터의 β-글루코시다아제 및 셀로덱스트린 운반체 NCU00801 를 포함하는 SL01 스트레인은 가장 높은 당 소비 및 에탄올 생산성을 나타내었다. 따라서, 이러한 스트레인을 추가적인 특성 해석을 위해 선택하였다.
SL01 및 SL00 둘 다를 진탕-플라스크 및 바이오리액터(bioreactors) 둘 다에서, 40 g/L 셀로비오스 및 50 g /L D-자일로스의 혼합물을 사용하여 배양하였다(도 66). 진탕-플라스크 배양에서(도 66A-B), 83 % 셀로비오스는 SL100(0.33 g/L/h 부터 0.46 g/L/h 까지)과 비교하여 평균 D-자일로스 소비율(consumption rate)이 41.2 % 보다 높으면서, SL01 에 의한 96 시간에 83 % 셀로비오스가 소비되었다. 증진된 당 소비율과 일치하는, 1.32-배의 증가된 평균 바이오매스 성장비율이 관찰되었다(0.031 g 의 세포건조무/L/h 내지 0.072 g 의 세포건조무/L/h 까지). 에탄올 생산성은 0.07 g/L/h 부터 0.23 g/L/h 까지 2.1-배 보다 증가되었다. 0.31 g/당 g (0.31 g per g sugar)의 가장 높은 에탄올 수득은 48 시간에 이르렀고, SL00 스트레인과 비교하여 23 % 의 증가를 나타낸, 평균 에탄올 수득은 0.28 g/당 g(0.28 g per g sugar)이었다. SL01 배양에서, 글루코스 및 D-자일로스의 공동-발효에서의 글루코스 억압(repression)의 특징(hallmark)인 유도기(lag phase)없이 보다 빠른 D-자일로스 소비율이 관찰되었다. 게다가, 증진된 바이오매스 성장(enhanced biomass growth) 및 에탄올 생산이 또한 관찰되었다.
Multifors system(스위스 보트민겐에 위치한 Infors-HT)을 바이오리액터에서 혼합된 당 발효를 위해 사용하였다. 각각의 용기(vessel)는 750 mL 의 전체 용량 부피를 가졌다. 각각의 용기에 대해서, p02 감지기(sensor), 공기 스파아저(air sparger), 출구 가스 냉각기(exit gas cooler), 온도 감지기, 배양 포트(inoculation port), 예비 부품(spare port), 침적관(dip tube), 소포 감지기(antifoam sensor), pH 감지기, 구동축(drive shaft), 블럭히터(heater block), 로터미터(rotameter), 연동 펌프 시스템(peristaltic pumps system)의 각각의 세트가 있다. 전체 바이오리액터 시스템은 냉각 시스템 ThermoFlex900(미국 매사추세츠주 월섬에 위치한 Thermo Scientific)가 갖추어져있다.
효모 스트레인의 단일 콜로니를 2 % 글루코스에 더하여 2 mL SC-Ura-Leu 배지에서 첫 번째로 성장시킨 다음에, 혼합된 당 발효 연구를 위해서 충분한 세포를 수득하기 위해 250 mL 의 진탕 플라스크에서 50 mL 의 동일한 배지 내에서 접종하였다. 하루 성장시킨 후에, 10 mL 의 포화 배양물을, 4 % 셀로비오스 및 5 % D-자일로스, 또는 4 % 셀로비오스, 5 % 자일로스, 및 0.5% 글루코스, 또는 4 % 셀로비오스, 5 % 자일로스, 및 1 % 글루코스로 보충된 400 mL 의 YPA 배지에 접종하였다. 온도를 30 ℃ 로 유지시켰고, pH 는 2 N H2SO4 또는 4 N NaOH 를 첨가하여 pH 5.5 를 유지시켰다. 처음 48 시에, 250 rpm 의 임펠러 속도(impeller speed)로 0.5 L/min 으로 송풍량(air flow rate)을 유지시켰다. 그 뒤에, 산소 제한 조건 하에서 높은 에탄올 생산을 달성하기 위해, 송풍량을 0.2 L/min 으로 조절하였다. 다양한 시점에 세 번의 샘플을 채취하였고, OD600, 당 농도 및 에탄올 농도를 상기한 바와 같이 결정하였다.
바이오리액터 배양에서(도 66C-D), 44 % 증가된 D-자일로스 소비율(0.47 g/L/h 에서 0.68 g/L/h 까지) 및 1.1-배 증가된 바이오매스 성장율(0.08 g 세포건조무/L/h 에서 0.17 g 세포건조무/L/h 까지)을 나타내면서, 48 시간 내에 대부분 모든 셀로비오스 및 66 % D-자일로스는 소비되었다. 에탄올 생산성은 4.3-배(0.09 g/L/h 에서 0.50 g/L/h 까지)보다 증가 되었고, 에탄올 수득은 0.39 g/당 g 이다. 진탕-플라스크 배양과 비교하여서, 처음 24 시간에서의 당 소비율은 낮았고, 이는 배치 배양의 시작에 사용된 낮은 세포 밀도 때문이다.
예상외로, 비록 발효에 글루코스를 첨가하지 않았을지라도, 작은 양의 글루코스가 발견되었다(도 66A-B). 최고의 글루코스 농도가 진탕-플라스크(12.1 g/L) 및 바이오리액터(bioreactors)(17.5 g/L) 둘 다에서 대략 24 시에 도달하였고, 그리고 난 다음에 매우 낮은 수준에 떨어졌다. 그러나 명백한 글루코스 억압이 이러한 글루코스의 존재 하에서 조차도 관찰되지 않았다. 글루코스가 SL00 스트레인에서 발견되지 않았기 때문에, β-글루코스가 이의 에피머 α-글루코스로 천천히 전환(slow conversion)되면서 세포외 글루코스가 발생할 수 있고, 글루코스의 주요한 형태(main form)는 해당작용(glycolysis)에서 사용되었다. 일반적으로, 글루코스에서 이의 상대적으로 낮은 농도 때문에, β-글루코스는 효소적으로(enzymatically) 또는 화학적으로(chemically) α-글루코스로 효율적으로 전환될 수 있다(Bouffard 등의, 1994). 그러나, 유전자 조작된 SL01 스트레인에서, β-글루코시다아제에 의한 촉매에 의해서, 과량의 β-글루코스는 세포 내에서 셀로비오스로부터 생산되었고, β-갈락토스와 함께 관찰된 것과 유사한, 작은 분획이 세포 바깥쪽으로 분비될 것이다(Bouffard 등의, 1994).
소량의 글루코스(전체 당의 10 % 미만)가 산업현장(industrial settings)에서 리그노셀룰로스계 가수분해물(lignocellulosic hydrolysates)에 일반적으로 존재하기 때문에, 유전자 조작된(engineered) SL01 스트레인의 발효 성과(fermentation performance)는 셀로비오스, D-자일로스 및 글루코스의 혼합물을 사용하여 조사하였다. 글루코스의 두 가지의 농도, 5 g/L 또는 10 g/L 를, 혼합된 탄소원으로서 40 g /L 셀로비오스 및 50 g/L D-자일로스와 함께 결합하였다. 5 g/L 글루코스(도 67A-B)를 사용하여, 67.8 % D-자일로스가 배치 배양(batch cultivation)에서 48 시간에 소비되면서, 81.7 % 의 셀로비오스를 SL01 에 의해 소비되었다. D-자일로스 소비율(consumption rate)은 0.32 g/L/h 부터 0.69 g/L/h 까지, 1.19-배 증가되었다. 에탄올 생산성은 3.3-배 증가된 반면에(0.11 g/L/h 부터 0.46 g/L/h 까지), 에탄올 수득은 0.26 g/g 당(g per g sugar) 부터 0.33 g/g 당으로 증가되었다. 10 g/L 글루코스(도 67C-D)를 사용하여, 74.7 % D-자일로스가 배치 배양(batch cultivation)에서 48 시간에 소비되면서, 83.8 % 의 셀로비오스를 SL01 에 의해 소비되었다. D-자일로스 소비율(consumption rate)은 0.45 g/L/h 부터 0.76 g/L/h 까지, 68 % 로 증가되었다. 에탄올 생산성은 2.1-배 증가되었고(0.16 g/L/h 부터 0.50 g/L/h 까지), 에탄올 수득은 0.30 g/g 당(g per g sugar) 부터 0.33 g/g 당으로 증가되었다. 예상대로, 유전자 조작된(engineered) SL01 스트레인은, SL00 야생형 스트레인 보다, 보다 높은 효율성의 당 소비 및 보다 높은 비율의 에탄올 생산을 나타내었다. 보다 중요하게, 글루코스가 전체 당의 10 % 까지와 함께, 세 가지 당의 공동-발효(co-fermentation)에서 현저한 글루코스 저해가 없었고(도 67C-D), 이는 이러한 접근이 산업에서의 적용(industrial applications)에 대해서 실행가능할 수 있음을 암시한다.
유사한 연구를, 세 가지 N. 크라사 셀로덱스트린 운반체 NCU00801, NCU08114 및 NCU00809 를 글루코시다아제 NCU00130 와 함께 도입된, S. 세레비시아 스트레인 D452-2 에서 실행하였다. 형질전환체(transformants)를, 세포내 β-글루코시다아제(NCU00130)가 발현된 20 g/리터 셀로비오스를 포함하는 YSC 배지에서 선별하였다. 이러한 작업에서 사용된 스트레인 및 플라스미드를 표 17 에 나타내었다(Ex. 12). 사용된 프라이머를 표 28 에 나타내었다.
[표 28]
각각 자일로스 또는 셀로비오스 발효 실험에 대해서 접종물(inoculums)을 제조하기 위해, 효모를 20 g/L 의 글루코스 또는 20 g/L 의 셀로비오스를 포함하는 YP 배지에서 성장시켰다. 20 g/L 의 글루코스 또는 셀로비오스를 포함하는 YP 배지로부터의 중기-대수증식기(mid-exponential phase)를 채취하였고, 멸균수(sterilized water)로 2 번 세척한 후에 접종하였다(inoculated). 모든 플라스크 발효 실험은, 산소 제한 조건 하에서 초기의 OD600 의 0.1 에서, 30 ℃ 에서 250 mL 의 플라스크에 40 g/L 또는 80 g/L 의 자일로스를 포함하는 YP 배지의 50 mL 를 사용하여 실시하였다. 바이오리액터 발효(Bioreactor fermentations)는, 산소 제한된 250 조건 하에서 200 rpm 의 교반 속도(agitation speed)로 30 ℃ 에서, Sixfors Bioreactors (Appropriate Technical Resources, Inc)를 사용한 적절한 양의 당을 포함하는 400 mL 의 YP 배지에서 실시하였다. 초기 세포 밀도(Initial cell densities)는 OD600 = 1.0 을 조절되었다.
세포 성장을 UV-가시광선 분광광도계(UV-visible Spectrophotometer)(미국 뉴욕주 서모에 위치한 Biomate 5)를 사용한 600 nm 에서의 광학 밀도(OD)로 모니터하였다. 글루코스, 자일로스, 자일리톨, 글리세롤, 아세테이트 및 에탄올 농도를, 265 a Rezex ROA-유기산 H+ (8%) 컬럼(265 a Rezex ROA-Organic Acid H+ (8%) column)(미국 캘리포니아주 토런스에 위치한 Phenomenex Inc.)을 사용한 시차 굴절률 검출기(refractive index detector)가 갖추어진 고성능 액체 크로마토그래피 265(HPLC, Agilent Technologies 1200 Series)로 결정하였다. 컬럼을 50 ℃ 에서 0.6 mL/min 의 유량(flow rate)으로 0.005 N 의 H2SO4 으로 용출시켰다.
셀로비오스가 단일 탄소원이면, 세 가지 모두의 형질전환체(transformants)는 에탄올을 성장하고 생산할 수 있지만(도 68), 세 가지의 형질 전환체는 상이한 셀로비오스 발효 비율을 나타내었다(NCU00801 > NCU08114 > NCU00809). NCU00801 및 NCU00130 둘 다가 발현된 가장 빠른 셀룰로스-발효 형질전환체(D801-130)는, 16.8 g/L 의 에탄올을 생산하면서, 4 시간 내에 40 g/L 의 셀로비오스를 소비하였다. 셀로비오스 발효의 부피 생산성(volumetric productivity)(P에탄올/ 셀로비오스 = 0.7 g/L/h)은 글루코스 발효(P에탄올/ 글루코스 = 1.2 g/L/h) 보다 낮았고, 셀로비오스로부터의 에탄올 수득(Y에탄올/ 셀로비오스 = 0.42 g/g)은 동일한 배양 조건 하에서, 글루코스로부터의 에탄올 수득(Y에탄올/ 글루코스 = 0.43 g/g)과 거의 같다. 그러나, D801-130 에 의한 관찰된 셀로비오스 소비율(cellobiose consumption rate) 및 에탄올 수득은, β-글루코시다아제의 표면 전시(surface display)를 통해 셀로비오스를 발효시키기 위해 유전자 조작된 S. 세레비시아 스트레인 상에서 개선되었다(Kotaka 등의, 2008; Nakamura 등의, 2008). 이러한 결과는, S. 세레비시아에서 NCU00801 및 NCU00130 의 동시 발현은 효율적인 셀로비오스 발효를 야기할 수 있음을 나타낸다.
효율적인 자일로스 발효 스트레인 DA24-16(실시예 13 에 기재)을 성장시킨 후에, 셀로덱스트린 운반체 및 β-글루코시다아제를 코딩하는 유전자(NCU00801 및 NCU00130) 효소를, 이것이 셀로비오스 및 자일로스를 동시에 소비할 수 있는 스트레인 내로 도입시켰다. 이는, 셀로비오스의 세포내 가수분해(intracellular hydrolysis)로 인하여, 자일로스 이용(xylose utilization)의 글루코스 저해는 이러한 스트레인에서 완화되는 가설을 세울 수 있다. NCU00801 유전자는 DA24-16 의 게놈(genome) 내로 통합되었고, NCU00130 은 다중-복제 플라스미드(multi-copy plasmid)로부터 발현되었다. 결과적으로 생성된 형질전환체, DA24-16-BT3 는 단일 탄소원(sole carbon source)으로서 셀로비오스를 함유하는 한천 평판(agar plate)에서 선별되었다.
다양한 양의 셀로비오스 및 자일로스를 포함하는 배지에서 성장한 DA24-16- BT3 스트레인은 셀로비오스 및 자일로스를 공동-소비하였고(co-consumed), 실험한 모든 조건에서 0.38-0.39 g/g 의 수득으로 에탄올을 생산하였다(도 69). 공동-발효의 잠재적인 상승 효과(potential synergistic effects)를, 세 가지의 상이한 조건 하에서 DA2416- BT3 를 배양하여 테스트하였다 : 40 g/L 의 셀로비오스, 40 g/L 의 자일로스, 및 40 g/L 의 둘 다의 당(총 80 g/L 의 당). 놀랍게도, DA24-16BT3 은, 별도로 40 g/L 의 셀로비오스 또는 40 g/L 의 자일로스가 소비되기 위해 필요로하는 동일한 기간 내의 80 g/L 의 셀로비오스/자일로스 혼합물을 공동-소비할 수 있었다(co-consume)(도 70). 게다가, DA24-16BT3 은, 단일 당 발효(셀로비오스 또는 자일로스)로부터의 에탄올 수득(0.31 ~ 0.33 g/g)과 비교하여, 셀로비오스 및 자일로스의 혼합물로부터 보다 높은 수득(0.39 g/g)을 갖는 에탄올을 생산하였다. 에탄올 생산성은 또한 공동-발효(co-fermentation) 동안에 0.27 g/L/h 에서 0.65 g/L/h 까지로 대폭 증가되었다. 셀로비오스 및 자일로스의 공동-발효(co-fermentation)는 전체적인 에탄올 수득 및 생산성을 향상시킬 수 있는, 이러한 결과를 나타내었다. 발효 실험(Fermentation experiments)은 셀로비오스 및 자일로스를 유효하게 공동-발효시킬 수 있는, P. 스티피티스와 이러한 유전자 조작된 S. 세레비시아 스트레인(DA24-16BT3)을 비교하기 위해 또한 실행하였다.
에너지케인(energycane)의 조성을 기반으로 한 모조의 가수분해물(simulated hydrolysate)(10 g/L 의 글루코스, 80 g/L 의 셀로비오스, 40 g/L 의 자일로스)를 사용하였다. 상이한 리그노셀룰로스계 식물(lignocellulosic plants)의 조성(composition)은 광범위한 범위로 다양하다. 예를 들어, 에너지 바이오매스 데이터베이스(Energy biomass database)의 US 부(US Department)는 150 이상의 바이오매스 샘플의 조성을 나타내었다(웹페이지 eere.energy.gov/biomass/rn/feedstock _databases.html). 이러한 샘플의 셀룰로스 대 헤미셀룰로스의 비율(cellulose-to-hemicellulose ratios)은 1.4 내지 19 사이이고, 평균은 2.3 이다. 에너지 작물(Energy crops)은 일반적으로 목질의 바이오매스(woody biomass) 보다 높은 헤미셀룰로스 함량(content)을 갖는다. 사탕수수 바가스(sugarcane bagasse), 옥수수 대(corn stover), 수수(sorghum)의 셀룰로스 대 헤미셀룰로스의 평균 비율은 각각 2.0, 1.85 및 2.14 이다. 따라서, 우리는 우리의 모조의 당 실험 설계(our simulated sugar experiment design)에서 2 의 글루칸/자일란 비율을 사용하였다. 유전자 조작된 효모는, 바이오연료 생산을 위한 β-글루코시다아제 활성(activities)에 결핍된, 전통적인 셀룰라아제 칵테일과 함께 사용될 것으로 예상될 것이다. 바이오매스 가수분해 과정은, 불완전한 셀룰라아제 칵테일(incomplete cellulase cocktails)을 갖는 6-30 % 의 글루칸-대-글루코스의 전환(glucan-to-glucose conversions)이 보고된 바와 같이, 리그노셀룰로스계 가수분해물에서 소량의 글루코스를 야기할 것이다(Medve 등의, 1998). 상기의 모든 요인을 고려하여, 10 g/L 글루코스, 80 g/L 셀로비오스 및 40 g/L 자일로스의 당 조합(combination)을 모조의 당 실험(simulated sugar experiments)에서 선택하였다.
DA24-16BT3 는 셀로비오스 및 자일로스가 순식간에 공동-소비(co-consuming)되기 전에 글루코스를 첫 번째로 소비하였다. 비록 작은 접종물(inoculums)을 사용하였을지라도(OD600 = 1), 총 130 g/L 의 당을 60 시간 내로 소비하였다. 이와 대조적으로, P. 스티피티스는 동일한 배양 조건 하에서 동일한 기간 내에 당 혼합물의 발효를 끝낼 수 없었다(도 71). DA24-16BT3 는 60 시간 내에 48 g/L 의 에탄올을 생산하였다(Y에탄올/당 = 0.37 g/g 및 P에탄올/당 = 0.79 g/L/h).
셀로비오스 소비 동안의 배지에서의 셀로덱스트린의 일시적인 축적(transient accumulation)이 관찰되었다(도 72~73). 축적된 셀로트리오스 및 셀로테트라오스는 셀로비오스의 고갈된 후에 다시 소비되었다. 축적된 셀로덱스트린은 β-글루코시다아제(NCU00130)의 트랜스-글리코실화 활성도(trans- glycosylation activity)(Christakopoulos 등의., 1994)에 의해 생성된 것 같고, 양 방향성(both directions)(세포내 <-> 세포외)으로 셀로덱스트린의 수송을 용이하게 할 수 있는, 셀로덱스트린 운반체(NCU00801)에 의해 분비되었다. 이러한 일시적인 셀로덱스트린의 축적은, 축적된 셀로덱스트린이 유전자 조작된 효모에 의해 결국에 소비되기 때문에, 생산 수확량(product yields)을 아마도 감소시키지 않을 것이다. 그러나, 셀로트리오스 및 셀로테트라오스의 수송 비율(transport rates)이 셀로비오스 보다 더 늦을 수 있기 때문에, 이는 생산성을 감소시킬 수 있다.
소량의 글루코스는 공동-발효(co-fermentation) 동안에 배지에서 끊임없이 발견되었다. 훨씬 작은 양의 글루코스 축적은 자일로스를 억제할 수 있기 때문에, 글루코스 수준을 최소한으로 유지시켜야한다. 셀로덱스트린 운반체 및 β-글루코시다아제의 상대적인 발현 수준은 글루코스의 축적에 영향을 미칠 가능성이 있음을 가설을 세울 수 있다. NCU00801 가 효모 게놈 내로 통합되었을때 보다, NCU00801 가 다중-복제 플라스미드(multi-copy plasmid)에 도입시켰을 때 배지에서 보다 많은 글루코스가 축적됨을 관찰된 사실이 이를 지지한다. 잠재적인 이유가 글루코스 저해인(potential reason being glucose repression), 다중-복제 플라스미드에서 NCU00801 및 NCU00130 둘 다를 함유하는 스트레인(DA24-16-BT)은, DA24-16-BT3 에서 관찰된 자일로스 이용 비율 보다, 상대적으로 더 느린 자일로스 이용 비율(xylose utilization rates)을 나타내었다(도 74). 셀로덱스트린 운반체 및 β-글루코시다아제 발현 수준의 추가적인 수정(adjustments) 또는 감소된 트랜스-글리코실화 활성도(trans-glycosylation activities)를 갖는 β-글루코시다아제의 확인은 공동-발효(co-fermentation) 동안에 글루코스 및 셀로덱스트린의 축적을 감소시킬 수 있다.
자일로스 및 셀로비오스의 공동-발효는 두 가지의 상이한 효모 스트레인의 혼합된 배양에 의해 또한 성취될 수 있다 : 자일로스-발효(fermenting) DA24-16 스트레인 및 셀로비오스-발효 DA452BT(도 75). 상기에 설명한 바와 같이, 효모 스트레인 DA24-16 는 자일로스-활용 효소 야생형 자일로스 환원효소(XYL1), 돌연변이체 자이로스 환원효소 R276H(mXYL1), 자일리톨 탈수소효소(XYL2) 및 자일루로키나아제(XKS1)가 발현되었다(Ex. 12; 표 17). D452BT 는, 셀로덱스트린 운반체 NCU00801 및 β-글루코시다아제 NCU00130 가 발현될 D452 가 유전자 조작되어 형성되었다. 혼합된 배양물에서, DA24-16 스트레인은 자일로스를 흡수하고(took up)(도 75A 에 녹색 오각형으로 나타낸 자일로스 분자), 효소 XYL1(야생형 및 돌연변이체), XYL2 및 XYL3 을 사용하여 이를 대사 작용하는 반면에, 그 밖의 스트레인 D452BT 는 운반체 NCU00801 를 사용하여 셀로비오스(도 75A 에 두 개의 빨간 색의 육각형으로서 나타낸 셀로비오스 분자)를 흡수할 수 있고, 효소 NCU00130 을 사용하여 셀로비오스를 글루코스로 전환된다. 이런 이유로, 혼합된 배양물은 에탄올을 생산하기 위해 자일로스 및 셀로비오스가 공동-발효될 수 있다(도 75 B).
이러한 연구는 S. 세레비시아에 의한 육탄당 및 오탄당 당의 공동-발효를 가능하게 하기 위한 새로운 전략을 보여준다. 셀로덱스트린 수송 시스템과 함께 효율적인 자일로스 활용 경로가 결합됨으로써, 글루코스 저해에 의해 초래되는 문제가 극복되었다. 결과적으로, 유전자 조작된 효모는 에탄올에 대한 셀룰로스의 가수 분해물 상승 작용(cellulosic hydrolysates synergistically)에 의해 두 가지의 비-대사 작용 가능한 당(two non-metabolizable sugars)을 공동-발효시켰다. 본원에 기재된 새로운 공동-발효 방법은 당화(saccharification) 및 발효 전선(fermentation fronts) 둘 다에서 리그노셀룰로스계 기술(lignocellulosic technologies)이 증진되었다. 대부분의 전통적인 균류에 의한 셀룰라아제 칵테일은 β-글루코시다아제가 부족하고, 효모에서 유효하게 발효되지 않는 셀로비오스와 함께 셀룰로스 가수분해(cellulose hydrolysis)를 끝낸다. 결과적으로, 추가의 β-글루코시다아제 효소는 셀로비오스를 글루코스로 전환시키기 위해 첨가해야한다. 셀로비오스/자일로스 공동-발효 효모는, 셀룰로스 당화 과정(cellulose saccharification process)과 관련된 에너지 사용(enzyme usage) 및 비용을 낮추면서 제한된 β-글루코시다아제 활성도를 갖는 이러한 셀룰라아제 칵테일(cellulase cocktails)의 사용을 가능하게 한다. 추가적으로, 셀로비오스 및 자일로스 공동 발효 사이의 시너지 효과(synergy)는 에탄올 생산성(ethanol productivity)을 현저하게 증가시키고, 따라서 발효의 경제성(fermentation economics)을 개선시킨다. 리그노셀룰로스계 물질(lignocellulosic materials)의 예비-처리 및 가수분해로부터 소량의 글루코스의 존재는, 육탄당 및 오탄당 당 혼합물을 에탄올로 전환시키기 위해 유전자 조작된 호모의 생산 능력에 영향을 주지 않는다.
이러한 연구는 식물 바이오매스(plant biomass)로부터 모방된 가수분해물(mimic hydrolysates)인 것으로 여겨지는 다양한 혼합물의 당을 발효시키는 유전자 조작된 S. 세레비시아 스트레인의 능력을 측정하는 것을 포함한다. 셀로덱스트린 및 자일로스의 공동-발효를 위한 이러한 스트레인의 능력은 예비-처리된 식물 바이오매스(pre-treated plant biomass)의 동시적인 당화 및 공동-발효(simultaneous saccharification and co- fermentation, SSCF) 동안에 특히 유용하다. SSCF 동안에, 헤미셀룰로스는 산 예비-처리에 의해 처음으로 가수분해되고, 이는 자일로스 및 여전히-결정질인 셀룰로스(still-crystalline cellulose)의 형성을 야기한다. 그리고 난 후에, 본원에 기재된 균류에 의한 셀룰라아제(fungal cellulases) 및 효모 스트레인이 첨가되고, 셀룰라아제는 자일로스 및 셀로비오스를 에탄올로 공동-전환되게 한다. 이러한 계획에서의 제한된 세포외 글루코스 생산 때문에, 자일로스 활용의 억제(repression)는 감소할 것이고, 공동-발효(co-fermentation)는 신속하고, 상승작용에 의해 진행될 것이다.
이러한 연구에서 사용된 S. 세레비시아는 실험실 스트레인(laboratory strain)일지라도, 발표된 결과(published results)와 비교하였을 때, 유전자 조작된 스트레인의 발효 수행(fermentation performance)은 매우 인상적이다. 중요한 발효 파라미터(수득 및 생산성)은 발판(platform)으로서의 산업적인 효모 스트레인의 용도에 의해 추가적으로 개선될 수 있다. 이러한 공동-발효 전략(co-fermentation strategy)의 응용(Applications)은 에탄올 생산에 제한될 수 있다. 이는 기초적인 기술이기 때문에, 본원에 나타낸 전략은, 화학적 동반물질(commodity chemicals) 및 증진된 바이오연료를 생산하기 위한, 어떠한 모든 제품 다양화 기술(any other product diversification technologies)과 함께 결합될 수 있다.
자일란에서
성장한 N.
크라사의
전사체
분석(
Transcriptome
Analysis
)
리그노셀룰로스계 바이오매스는 셀룰로스, 헤미셀룰로스 및 리그닌(lignin)으로 구성되었다. 실시예 1-3 은 N. 크라사의 전사체(transcriptome) 및 세크레톰(secretome) 분석을 통해 셀룰로스에서의 성장에 중요한 유전자의 발견을 기재하였다. 이러한 실시예에서, N. 크라사 게놈의 발현 프로파일(expression profile)은, 헤미셀룰로스의 활용에 중요한 유전자를 결정하기 위해 자일란에서의 성장 동안을 조사하였다.
WT 또는 △ xlnR 스트레인의 10 일 된 분생자(Ten day old conidia)는, 100 mL 의 IX Vogel's 염 최소 배지(IX Vogel's salts minimal medium)(2 % 글루코스)에서 106 분생자/mL 에 접종하고, 일정한 빛(constant light)으로 25 ℃ 에서 16 시간 성장시키고, IX Vogel's 배지만(IX Vogel's only medium)으로 세척하였다. 분생자는 배지에서 단일 탄소원(sole carbon source)으로서 2 % 글루코스 또는 2 % Beechwood 자일란을 갖는 lOOmL IX Vogel's 염 내로 이동시킨 다음에, 4 시간 동안 성장시켰다. 균사체(Mycelia)를 여과에 의해 채취하였고, 액체 질소에서 즉시 급속 냉동시켰다(flash frozen). 전체 RNA 를 제조자의 지시에 따라 TRIzol(Invitrogen)을 이용하여 분리하였고, DNase 로 처리하였다(Turbo DNA-free 키트 ; Ambion) (Kasuga, Townsend 등의, 2005).
cDNA 합성 및 표시(labeling)를 위해, Pronto kit(카달로그 번호 제 40076 호 ; Corning)를 사용된 총 RNA 가 10 ㎍ 당 샘플인 점을 제외하고는 제조자의 상세한 설명(specifications)에 따라 사용하였다.
마이크로어레이 혼성(Microarray hybridization) 및 데이터 분석을 사전에 기재한 바와 같이 실시하였다(Tian, Kasuga 등의, 2007). GenePix 4000B 스캐너(scanner)(Axon Instruments)을 사용하여 이미지(images)를 획득하였고, GenePix Pro6 소프트웨어를 사용하여 혼성 신호(hybridization signals)를 정량하고 미가공 데이터(raw data)를 수집하였다. 정규화된 발현 값(Normalized expression values)은 BAGEL(Bayesian analysis of gene expression levels : 유전자 발현 수준의 베이지안 분석) 스프트웨어 프로그램을 사용하여 분석하였다(Townsend and Hartl 2002; Townsend 2004). 354 유전자는 자일란 상에서 성장한 N. 크라사에서 2-배 보다 더 유도된 점이 발견되었다. 이러한 목록을 도 76 에 나타내었다.
자일란에서
성장한 N.
크라사의
세크레톰
분석
자일란에서 성장하는 동안의 N. 크라사의 세크레톰(secretome)은 샷건 프로테이믹스 접근법(shotgun proteomics approach)을 사용하여 분석하였다. 자일란 배양물로부터의 상청액(Supernatants)을 트림신으로 소화시키고, 액체 크로마토그래피 나노-전자분사 이온화 이중 질량분석기(nano-electrospray ionization tandem mass spectrometry)로 분석하였다.
질량 분석 샘플(Mass spectrometry samples)을 하기와 같이 제조하였다. N. 크라사 야생형 스트레인은 4 일 또는 7 일 동안 2 % 자일란 배지에서 성장시켰다. 배양 상청액을 원심분리로 분리하고, 0.22 ㎛ 필터를 통해 여과시키고, 10 kDa MWCO PES 스핀 농축기(spin concentrators)로 10 번 농축시켰다. 3.36 mg 의 요소, 5 μL 의 1 M 트리스 pH 8.5 및 5 μL 의 100 mM DTT 를 100 μL 의 농축된 배양 상청액에 더하고, 혼합물을 1 시간 동안 60 ℃ 로 가열하였다. 가열 후에, 700 μL 의 25 mM 중탄산 암모늄 및 140 μL 의 메탄올을 용액에 첨가한 다음에 pH 5.0 의 50 mM 아세트산 나트륨에 50 μL 의 100 ㎍/mL 트립신으로 처리하였다. 트립신을, 기저(basal) pH 에서 약 8-9 시간 동안 인버팅(inverting)하면서 37 ℃ 에서 밤새 반응시켰다. speedvac 으로 건조시키기 위해 소화 부피(digestion the volume)를 감소시킨 후에, 300 μL MilliQ 물로 3 번 세척하였다. 최종적인 부피는 100 μl 이다. TFA 를 0.1 - 0.3 % v/v 으로 첨가하였다. 샘플에서의 잔여 염(Residual salts)을 제조자의 지시에 따라 OMIX 마이크로 추출 피펫 팁(microextraction pipette tips)을 사용하여 제거하였다. 아세토니트릴을 증발(evaporation)로 제거하였다. 샘플 용액은 0.1 % - 1 % TFA 을 갖는 수용액(aqueous solution)이고, 최종 부피는 10 마이크로리터 이상이다.
분비된 단백질(
secreted
proteins
)을
코드하는
것으로 예측된
자일란
-유도된 유전자(
xylan
-
induced
genes
)의 분석
전사체 및 세크레톰 분석은 나타낸 전체의 71 가지의 유전자 중에서 55 가지가 분비되는 것으로 예측된 결과를 나타내었다. 이러한 유전자를 표 29 에 나타내었다. 결실 스트레인(deletion strains)은 69 가지 유전자 중에 49 가지의 유전자에 대해서 이용할 수 있다. 이러한 46 가지의 유전자 중에서, 6 가지의 스트레인은 이핵 접합체(heterokaryons)이고, 그 외의 남아있는 40 가지의 결실 스트레인은 전체 분비된 단백질, 존재하는 자일로스의 양 및 아조-엔도-자일라나아제 활성도(azo-endo-xylanase activity)에 대해서 분석하였다. 결과를 도 77 에 나타내었다.
[표 29]
샘플을 하기와 같이 제조하였다. 10 일 된 분생자(10 day old conidia)를 106 분생자/mL 의 100 mL 의 2 % 자일란 Vogel's 배지에서 성장시켰다. 두 가지의 복제물(replicate)을 각각의 스트레인에 대해서 제조하였다. 배양물을 일정한 빛(constant light) 및 220 rpm 으로 25 ℃ 에서 성장시켰다. 샘플을 4 일에 채취하였고, 상청액을 원심분리로 분리하고 검정에서 사용하였다.
브레드포드 단백질 농도(Bradford protein concentrations)를, 분비된 단백질의 총량을 결정하기 위해 측정하였다. 스톡(Stocks)을 BSA 표준(BSA standards)으로 제조하였다 : 0 ㎍/mL, 50 ㎍/mL, 100 ㎍/mL, 250 ㎍/mL 및 500 ㎍/mL. 브레드포드 용액을 1 : 4 로 희석하였다. 멀티 채널 피펫(multichannel pipette)을 96 웰 플레이트 내로 200 μL 의 브레드포드 용액을 피펫으로 재서 옮기기 위해(pipette) 사용하였다. 10 μL 의 샘플 및 10 μL 의 각각의 표준을 첨가하였다. 샘플을 10 분 동안 실온에서 배양하였다. 흡광도를 595 nm 에서 판독하였고, 단백질 농도를 결정하였다.
자일로스를 측정하기 위해 사용된 검정은 Bailey 등의, 1992 (J Biotech 23: 257- 270)로부터 수정되었다. 자일로스 표준(Xylose standards)은 H20 에서 제조되었다. 농축된 0.8 M 자일로스(10 mL 에서의 1.2 g)에 대해서, 표준은 0 mM, 8 mM(1 : 100 희석 ; 990 μl + 10 μl), 20 mM(1 : 100 희석 ; 975 μl + 25 μl), 40 mM(1 : 100 희석 ; 950 μl + 50 μl), 80 mM(1 : 100 희석 ; 900 μl + 100 μl) 및 160 mM(1 : 100 희석 ; 800 μl + 200 μl). 멀티 채널 피펫(multichannel pipette)을, 딥 웰 96-웰 플레이트(deep well 96-well plate)에 900 μL 의 기질 용액(substrate solution)을 첨가하는데 사용하였다. 기질을 10 분 동안 50 ℃ 에서 배양시켰다. 100 μL 의 배양 상청액 및 표준을 첨가하고, 5 분 동안 50 ℃ 에서 배양시켰다. 샘플을 10 분 동안 3,400 rpm 으로 원심분리하였다. 멀티 채널 피펫을, 96-웰 PCR 플레이트 내로 75 μL DNS 용액을 피펫으로 재서 옮기는데(pipette) 사용하였다. 5 μL 의 용액을 반응물로부터 제거하였고, DNS 용액을 포함하는 PCR 플레이트에 첨가하였다. 플레이트를 5 분 동안 PCR 기계에서 99 ℃ 로 가열하였다. 샘플을 냉각 시킨 후에, 이들을 확실히 바닥이 평평한 플레이트(flat-bottomed plates)에 이동시키고, 흡광도를 540 nm 에서 판독하였다. 기질 용액(500 mL)은 beechwood 자일란(5 g; 10 mg/mL), 3M NaOAc, pH 5.0 (8.33 mL; 50 mM) 및 물 (491 mL)을 포함하고, 이를 20 분 동안 오토클레이브하였다(autoclave). DNS 용액(100 mL)은 3, 5-디니트로살리실산(707 mg), NaOH (1.32 g), Rochelle 염[Na K 주석산염(tartrate)](20.4 g), 소듐 메타-바이설페이트(Sodium meta-bisulfate)(553 mg), 페놀(507 μL) 및 물 (94.4 mL)을 포함한다.
아조-엔도-자일라나아제 활성도(Azo-endo-xylanase activity)를 Megazyme 으로부터의 키트로 측정하였다. 이러한 검정을, 염료와 복합된 자일란 사슬(xylan chain)로부터의 해방된(liberated) 염료의 양을 분광광도계적으로(spectrophotometrically) 측정한 샘플에서의 엔도-자일라나아제 활성(endo-xylanase activity) 양을 간접적으로 측정하였다. 보다 많은 효소가 존재할수록, 보다 많은 염료가 방출될 것이다. 모든 상청액 샘플을, 분리된 15 mL 의 Falcon 튜브에서 450 μL 의 Na 아세테이트 완충용액(50 mM, pH 4.5)에 50 μL 의 상청액을 첨가하여 1 : 10 으로 희석하였다. 그 다음에, Falcon 튜브를 약 10 분 동안 예비-항온시켰다(pre-warmed). 기질 용액을 모든 튜브에 대한 모든 샘플(500 μL/샘플)에 첨가하였다. 샘플 및 기질 용액을 이들을 예비-평형시키기 위해(pre-equilibrate) 10 분 동안 40 ℃ 의 워터 배스(water bath)에 두었다. 500 μL 의 기질 용액을 각각의 1 : 10 희석된 샘플에 첨가하고, 10 분 동안 보텍스하고(vortexed), 10 분 동안 40 ℃ 에서 배양하였다. 각각의 샘플에 대해서 2.5 mL 의 침전 용액(precipitant solution)(95 % 에탄올)을 첨가하여 반응을 종료하였다. 튜브를 10 분 동안 실온에서 두었다. 튜브를 10 분 동안 보텍스하고(vortexed), 실온에서 10 분 동안 1,000 g 으로 원심분리하였다. 각각의 튜브로부터의 1 mL 의 상청액 용액을 큐벳트(cuvette) 내로 직접적으로 두고, 흡광도를 590 nm 에서 측정하였다. 이러한 공정(procedure)에 대해서 사용된 블랭크(blank)는 2.5 mL 의 침전 용액에 첨가된 500 μL 의 기질 용액으로부터의 상청액이다.
마지막으로, N. 크라사에서 헤미셀룰로스의 저하(degradation)에 영향을 미치는 것으로 본원에서 확인된 유전자의 변형물(modulation)은, 헤미셀룰로스와 같은 식물 세포 벽 성분에서의 성장 및 식물 세포 벽을 분해하는 능력을 증진시키는 유전자 조작된 스트레인을 용이하게 할 수 있는 것으로 예상된다. 관심있는 유전자(Genes of interest)는 예측된 글루카밀라아제(glucamylase)를 코드하는 NCU01517 ; 예측된 아라비노푸라노시다아제(arabinofuranosidase)를 코드하는 NCU02343 ; 보존된 가설단백질(conserved hypothetical protein)을 코드하는 NCU05137 ; 예측된 아세틸자일란 에스테라아제 전구체(predicted acetylxylan esterase precursor)를 코드하는 NCU05159 ; 보존된 가설단백질을 코드하는 NCU09133 ; 및 가설 단백질을 코드하는 NCU10040 을 포함한다.
헤미셀룰로스에서 세포의 성장은 NCU01517, NCU09133 또는 NCU10040 에 의해 코드된 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포를 제공하여서 증가될 수 있다. 숙주 세포는, 재조합 폴리뉴클레오티드가 발현된 것과 같은 헤미셀룰로스를 포함하는 배지에서 배양될 것이다. 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포 보다는 이러한 배지에서 보다 빠른 비율로 성장할 것이다.
△
NCU05137
스트레인의 추가적인 분석
실시예 1-3 및 18-20 에 기재된 바와 같이, NCU05137 은, 미스칸터스, 아비셀 또는 자일란 중 어떠한 것에서 N. 크라사의 성장 동안에 과잉발현된(overexpressed) 예측된 분비된 단백질(predicted secreted protein)이다. 아비셀에서 성장한 NCU05137 이 결핍된 N. 크라사의 결실 스트레인(deletion strain)은 증가된 엔도글루카나아제(endoglucanase), β-글루코시다아제 및 아비셀라아제 활성도(Avicelase activity)를 나타내었다. 자일란에서 성장한 NCU05137 결실 스트레인은 증가된 아조-엔도-자일라나아제 활성도(azo-endo-xylanase activity)를 나타내었다. 이러한 실시예에서 기재된 바와 같이, △NCU05137 의 상보성(complementation)은, NCU05137 유전자의 손실(loss)로 인한 △NCU05137 에 나타낸 표현형(phenotypes)의 다양성을 위해서 수행되었다.
ccgl 프로모터의 조절 하에서 C-말단 GFP 태그(tag)를 갖는 NCU05137 를 포함하는 플라스미드를 생성하였다. N. 크라사 분생자는 NCU05137-GFP 구조물(construct)과 함께 형질전환되었다. 실험을 표준 네우로스포라 절차에 따라 실시하였다(웹페이지 fgsc.net/Neuro spora/Neuro sporaProtocolGuide.htm).
야생형, △NCU05137 및 △NCU05137-NCU05137-GFP 스트레인의 전체 분비된 단백질 및 카르복시메틸 셀룰라아제(carboxymethyl cellulase, CMC) 활성도를 측정하였다. 각각의 스트레인의 배양물로부터의 100 μL 의 상청액을 채취하고, 이를 900 μL 의 브레드포드 염료(Bradford Dye)에 첨가하고, 595 nm 의 흡광도로 측정하여, 전체 분비된 단백질을 측정하였다. CMC 활성도(CMC activity)를, 각각의 스트레인 배양물 및 아조-CMD 키트(azo-CMC kit)(Megazyme SCMCL)로부터의 20 X 희석된 상청액으로 측정하였다. △NCU05137 넉아웃 스트레인(knockout strains)은 분비된 단백질 및 CMC 활성도의 증가된 수준을 나타내었다. △NCU05137 스트레인 내로의 GFP-태그된 NCU05137 의 도입(Introduction)은 야생형 수준으로 다시 돌아가(back to) 이러한 수준을 감소시켰다(도 78).
게다가, 상보적인 스트레인에서 NCU05137-GFP 의 위치(localization)를 관찰하였다. NCU05137-GFP 는 분생자 및 균사의 팁(hypha tip)의 세포 벽에 위치한다(도 79-80). 이러한 데이터는 GFP-태그된 NCU05137 단백질이 완전히 기능적이고, 이는 이러한 단백질의 생화학적인 활성도를 다루는 정제 및 실험에 사용될 수 있음을 나타낸다.
따라서, NCU05137 의 정상적인 기능(normal function)은 셀룰라아제 및 헤미셀룰라아제 유전자 발현의 도입과 관련된 신호 전달 과정(signaling processes)을 저해할 수 있다. 세포에서의 NCU05137 또는 NCU05137 의 상동(homolog)의 발현의 저해는 저 세포에서의 셀룰라아제 및 헤미셀룰라아제 활성도를 증가시키는 것 같고, 그 결과로 셀룰로스 또는 헤미셀룰로스의 성장을 증가시키는 것 같다. 셀룰로스 또는 헤미셀룰로스에서의 세포의 성장을, NCU05137 을 코드하는 폴리펩티드를 코드하는 내인성 폴리펩티드(endogenous polynucleotide)를 포함하는 숙주 세포를 제공하여 증가시킬 수 있다. 내인성 폴리펩티드의 발현은 저해될 수 있고, 세포는 셀룰로스 및/또는 헤미셀룰로스를 포함하는 배지에서 배양될 수 있다. 숙주 세포는, 내인성 폴리펩티드의 발현을 저해하지 않는 배지에서의 세포 보다, 상기 배지에서 더 빠른 비율로 성장할 것이다.
NCU07705 의
추가적인 분석
NCU07705 의 발현이 셀룰로스에서 N. 크라사의 성장 동안에 상향조절될 수 있음을(upregulated) 발견하였다. NCU07705 를 코드하는 폴리펩티드의 BLAST 분석은, 폴리펩티드가 전사 인자(transcription factors)를 포함하는 많은 C6 징크 핑거 도메인(zinc finger domain)과 높은 유사성(similarity)을 갖는 것으로 나타났다(도 1). 셀룰로스의 이용에서 NCU07705 의 역할을 추가적으로 조사하기 위해, NCU07705 가 결핍된 결실 스트레인(deletion strain)의 표현형(phenotype)은 평가되었다.
△NCU07705 스트레인은 2 % 셀룰로스(아비셀), PASC 또는 단일 탄소원으로서 CMC 에서 성장할 수 없지만(표 30), 수크로스, 자일란 및 자일로스에서 야생형 스트레인에 대해서는 유사한 속도(kinetics)로 성장하였다. NCU07705 가 셀룰라아제의 발현을 조절하는 역할을 하는지 여부를 결정하기 위해, 셀룰라아제 및 헤미셀룰라아제 유전자의 발현을, 셀룰로스에서 △NCU07705 의 성장 동안에 조사하였다. 야생형으로부터의 10 일 된 분생자(FGSC 2489) 및 △NCU07705 스트레인을 Vogel's 액체 MM(2 % 수크로스)(Vogel 1956) 내에 배양시키고 16 시간 동안 성장시켰다. 균사체(Mycelia)를 원심분리하고, l x Vogel's 염으로 세척하고 난 다음에, 2 % 수크로스 또는 2 % 아비셀을 갖는 Vogel's 배지 내에 이동시키고, 4 시간 동안 일정한 빛(constant light)에서 성장시켰다. 이들은 여과에 의해 채취하였고, 액체 질소에서 즉시 냉동시켰다. 전체 RNA 를 제조자의 지시에 따라 TRIzol(미국 캘리포니아주 칼즈배드에 위치한 Invitrogen)를 사용하여 분리하였고, DNase(Turbo DNA-free kit, 미국 캘리포니아주 포스터시티에 위치한 Ambion/Applied Biosystems)로 처리하였다(Kasuga 등의, 2005). ChipShotTM 간접적으로 라벨된/클린-업 시스템(Indirect Labeling/Clean-Up System)(카달로그 번호 제 Z4000 호, 미국 위스콘신주 매디슨에 위치한 Promega) 및 CyDye 포스트-라벨된 반응성 염료 팩(Post-Labeling Reactive Dye Pack)(카달로그 번호 제 RPN5661 호, 미국 뉴저지주 피스카타웨이에 위치한 GE Healthcare)를, RNA 의 양이 10 ㎍ 을 사용하는 것을 제외한 제조자의 지시에 따라 cDNA 를 합성 및 표지하기(label) 위해 사용하였다. 프로토(Pronto)! 혼성 키트(Hybridization Kit)(카달로그 번호 제 40076 호, 미국 매사추세츠주 로웰에 위치한 Corning)를 제조자의 상세한 설명에 따라 마이크로어레이 혼성화(microarray hybridization)를 위해 사용하였다.
데이터 분석을 사전에 기재된 바와 같이 실시하였다(Tian 등의., 2007). GenePix 4000B 스캐너(미국 캘리포니아주 유니언 시티에 위치한 Axon Instruments)를 사용하여 이미지를 획득하였고, GenePix Pro6 소프트웨어를 혼성 신호(hybridization signals)를 정량하고, 미가공된 데이터(raw data)를 수집하기 위해 사용하였다. 정규화된 발현 값을 BAGEL(Bayesian Analysis of Gene Expression Levels : 유전자 발현 수준의 베이지안 분석)을 사용하여 분석하였다(Townsend and Hartl, 2002). 예측된 셀룰라아제 유전자 중 어떠한 것도 △NCU07705 스트레인에 유도되지 않은 반면에, 예측된 헤미셀룰라아제 유전자의 도입(induction)은 영향받지 않았다(unaffected)(하기의 표 30 참조). 따라서, NCU07705 는 cdr -1, 셀룰로스 분해 조절인자 1(cellulose degradation regulator 1)로 명명되었다.
따라서, 셀룰로스에서의 세포의 성장은, NCU07705 에 의해 코드된 폴리펩티드를 코드하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포의 제공에 의해 증가될 것이다. 숙주 세포는, 재조합 폴리뉴클레오티드가 발현된 것과 같이 셀룰로스를 포함하는 배지에서 배양될 수 있다. 숙주 세포는, 재조합 폴리뉴클레오티드를 포함하지 않는 배지에서의 세포보다, 상기 배지에서 더 빠른 비율로 성장할 것이다.
[표 30]
SEQUENCE LISTING
<110> The Regents of the University of California
The Board of Trustees of the University of Illinois
British Petroleum Technology Ventures, Inc.
GLASS, N. Louise et al.
<120> METHODS AND COMPOSITIONS FOR IMPROVING SUGAR TRANSPORT,
MIXED SUGAR FERMENTATION, AND PRODUCTION OF BIOFUELS
<130> 67779-2000140
<140> Not Yet Assigned
<141> Concurrently Herewith
<150> US 61/285,526
<151> 2009-12-10
<150> US 61/271,833
<151> 2009-07-24
<160> 97
<170> FastSEQ for Windows Version 4.0
<210> 1
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> Sequence motif
<221> VARIANT
<222> 1
<223> Xaa = Leu or Ile or Val or Met
<221> VARIANT
<222> 3
<223> Xaa = Phe or Leu
<221> VARIANT
<222> 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16
<223> Xaa = Any Amino Acid
<221> VARIANT
<222> 17
<223> Xaa = Tyr or Phe
<400> 1
Xaa Tyr Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10 15
Xaa Asp
<210> 2
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Sequence motif
<221> VARIANT
<222> 1
<223> Xaa = Tyr or Phe
<221> VARIANT
<222> 2, 3, 5, 6, 7, 8, 9, 11, 12, 13, 14, 15, 16
<223> Xaa = Any Amino Acid
<221> VARIANT
<222> 10
<223> Xaa = Pro or Val or Phe
<221> VARIANT
<222> 17
<223> Xaa = Asp or Gln
<400> 2
Xaa Xaa Xaa Gly Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10 15
Xaa
<210> 3
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Sequence motif
<221> VARIANT
<222> 3
<223> Xaa = Arg or Lys
<400> 3
Gly Arg Xaa
1
<210> 4
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Sequence motif
<221> VARIANT
<222> 2, 3, 4, 5, 6, 7
<223> Xaa = Any Amino Acid
<221> VARIANT
<222> 8
<223> Xaa = Tyr or Phe
<400> 4
Arg Xaa Xaa Xaa Xaa Xaa Xaa Xaa Asn
1 5
<210> 5
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Sequence motif
<221> VARIANT
<222> 3
<223> Xaa = Ile or Val or Leu or Ala
<221> VARIANT
<222> 5, 6, 7
<223> Xaa = Any Amino Acid
<400> 5
Trp Arg Xaa Pro Xaa Xaa Xaa Gln
1 5
<210> 6
<211> 24
<212> PRT
<213> Artificial Sequence
<220>
<223> Sequence motif
<221> VARIANT
<222> 6, 8, 9, 10, 11, 12, 13, 14, 15, 17, 18, 19, 21, 22
<223> Xaa = Any Amino Acid
<400> 6
Pro Glu Ser Pro Arg Xaa Leu Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Ala
1 5 10 15
Xaa Xaa Xaa Leu Xaa Xaa Tyr His
20
<210> 7
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Sequence motif
<221> VARIANT
<222> 2
<223> Xaa = Gly or Ser or Thr
<221> VARIANT
<222> 4, 8
<223> Xaa = Any Amino Acid
<221> VARIANT
<222> 9
<223> Xaa = Leu or Ile or Val
<400> 7
Phe Xaa Gln Xaa Ser Gly Asn Xaa Xaa
1 5
<210> 8
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> Sequence motif
<221> VARIANT
<222> 2, 3, 4, 6, 7, 9, 11, 12, 13, 14
<223> Xaa = Any Amino Acid
<221> VARIANT
<222> 5
<223> Xaa = Tyr or Ile or Val
<221> VARIANT
<222> 16
<223> Xaa = Gly or Ala
<400> 8
Leu Xaa Xaa Xaa Xaa Xaa Xaa Glu Xaa Leu Xaa Xaa Xaa Xaa Arg Xaa
1 5 10 15
Lys Gly
<210> 9
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> Transmembrane helix
<400> 9
Pro Glu Ser Pro Arg
1 5
<210> 10
<211> 4
<212> PRT
<213> Artificial Sequence
<220>
<223> Transmembrane helix
<400> 10
Pro Glu Thr Lys
1
<210> 11
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> Sequence motif
<221> VARIANT
<222> 1
<223> Xaa = Leu or Ile or Val or Met or Ser or Thr or Ala or Gly
<221> VARIANT
<222> 2
<223> Xaa = Leu or Ile or Val or Met or Phe or Ser or Ala or Gly
<221> VARIANT
<222> 3
<223> Xaa = Any Amino Acid But Ser or His
<221> VARIANT
<222> 4
<223> Xaa = Any Amino Acid But Arg or Asp or Glu
<221> VARIANT
<222> 5
<223> Xaa = Leu or Ile or Val or Met or Ser or Ala
<221> VARIANT
<222> 6
<223> Xaa = Asp or Glu
<221> VARIANT
<222> 7
<223> Xaa = Any Amino Acid But Thr or Asp
<221> VARIANT
<222> 8
<223> Xaa = Leu or Ile or Val or Met or Phe or Tyr or Trp or Ala
<221> VARIANT
<222> 11
<223> Xaa = Arg or Lys
<221> VARIANT
<222> 12, 13, 14, 15, 16, 17
<223> Xaa = Any Amino Acid and up to two of them can be
present or absent
<221> VARIANT
<222> 18
<223> Xaa = Gly or Ser or Thr or Ala
<400> 11
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Gly Arg Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10 15
Xaa Xaa
<210> 12
<211> 26
<212> PRT
<213> Artificial Sequence
<220>
<223> Sequence motif
<221> VARIANT
<222> 1
<223> Xaa = Leu or Ile or Val or Met or Phe
<221> VARIANT
<222> 2, 6, 9, 10, 11, 12, 13, 14, 15, 17, 18, 20, 21, 22, 23, 24, 25
<223> Xaa = Any Amino Acid
<221> VARIANT
<222> 4
<223> Xaa = Leu or Ile or Val or Met or Phe or Ala
<221> VARIANT
<222> 5
<223> Xaa = Any Amino Acid But Val
<221> VARIANT
<222> 8
<223> Xaa = Any Amino Acid But Lys or Pro
<221> VARIANT
<222> 16
<223> Xaa = Leu or Ile or Phe or Tyr
<221> VARIANT
<222> 19
<223> Xaa = Glu or Gln
<221> VARIANT
<222> 26
<223> Xaa = Arg or Lys
<400> 12
Xaa Xaa Gly Xaa Xaa Xaa Gly Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10 15
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
20 25
<210> 13
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<221> VARIANT
<222> 1
<223> Xaa = Ala or Cys
<221> VARIANT
<222> 2, 4, 5, 6, 7
<223> Xaa = Any Amino Acid
<221> VARIANT
<222> 8
<223> Xaa = Any Amino Acid But Glu or Asp
<400> 13
Xaa Xaa Val Xaa Xaa Xaa Xaa Xaa
1 5
<210> 14
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Sequence motif
<220>
<221> VARIANT
<222> 1
<223> Xaa = Leu or Ile or Val or Met or Phe or Ser or Thr or Cys
<220>
<221> VARIANT
<222> 2
<223> Xaa = Leu or Ile or Val or Phe or Tyr or Ser
<220>
<221> VARIANT
<222> 3
<223> Xaa = Leu or Ile or Val
<220>
<221> VARIANT
<222> 4
<223> Xaa = Leu or Ile or Val or Met or Ser or Thr
<220>
<221> VARIANT
<222> 8
<223> Xaa = Leu or Ile or Val or Met or Phe or Ala or Arg
<220>
<221> VARIANT
<222> 9
<223> Xaa = Cys or Ser or Ala or Gly or Asn
<400> 14
Xaa Xaa Xaa Xaa Glu Asn Gly Xaa Xaa
1 5
<210> 15
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> Sequence motif
<220>
<221> VARIANT
<222> 1, 2
<223> Xaa = Leu or Ile or Val or Met
<220>
<221> VARIANT
<222> 3
<223> Xaa = Lys or Arg
<220>
<221> VARIANT
<222> 4, 6, 7, 8, 9, 16, 17
<223> Xaa = Any Amino Acid
<220>
<221> VARIANT
<222> 5
<223> Xaa = Glu or Gln or Lys or Arg or Asp
<220>
<221> VARIANT
<222> 11
<223> Xaa = Leu or Ile or Val or Met or Phe or Thr or Cys
<220>
<221> VARIANT
<222> 12
<223> Xaa = Leu or Ile or Val or Thr
<220>
<221> VARIANT
<222> 13
<223> Xaa = Leu or Ile or Val or Met or Phe
<220>
<221> VARIANT
<222> 14
<223> Xaa = Ser or Thr
<220>
<221> VARIANT
<222> 18
<223> Xaa = Ser or Gly or Ala or Asp or Asn or Ile or Thr
<400> 15
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Gly Xaa Xaa Xaa Xaa Asp Xaa
1 5 10 15
Xaa Xaa
<210> 16
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 16
atctgggaag cgaacaaag 19
<210> 17
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 17
tagcggtcgt cggaatag 18
<210> 18
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 18
cccatcacca ctactacc 18
<210> 19
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 19
ccagccctga acaccaag 18
<210> 20
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 20
tgatcttacc gactacct 18
<210> 21
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 21
cagagcttct ccttgatg 18
<210> 22
<211> 533
<212> PRT
<213> Sporotrichum thermophile
<400> 22
Met Gly Leu Ser Thr Lys Ile Leu Gln Lys Ile Val Arg Asn Glu Ala
1 5 10 15
Met Ala Ser Asp Pro Pro Glu Ile Tyr Gly Trp Arg Val Tyr Leu Leu
20 25 30
Ala Cys Ser Ala Cys Phe Gly Ala Met Ser Phe Gly Trp Asp Ser Ser
35 40 45
Val Ile Gly Gly Val Ile Val Leu Pro Pro Phe Ile Arg Asp Phe Asn
50 55 60
Leu Gly Asp Pro Lys Ser Gln Ala Ser Ala Asn Leu Ser Ala Asn Ile
65 70 75 80
Val Ser Thr Leu Gln Ala Gly Cys Phe Leu Gly Ala Leu Val Ala Ser
85 90 95
Pro Met Thr Asp Arg Phe Gly Arg Lys Trp Cys Leu Ile Gly Val Ser
100 105 110
Leu Ile Ile Ile Ile Gly Ile Ile Met Gln Ala Ala Ala Ser Gly Asn
115 120 125
Leu Gly Pro Met Tyr Ala Gly Arg Phe Ile Ala Gly Ala Gly Val Gly
130 135 140
Ala Ala Ser Thr Ile Asn Pro Ile Tyr Val Ser Glu Asn Ala Pro Arg
145 150 155 160
Ala Ile Arg Gly Leu Leu Thr Gly Leu Tyr Gln Leu Phe Ile Val Thr
165 170 175
Gly Gly Met Ile Ala Phe Trp Ile Asn Tyr Ser Val Ser Ile His Phe
180 185 190
Pro Glu Thr Lys Ile Met Tyr Val Phe Pro Leu Ala Ile Gln Ala Leu
195 200 205
Pro Ala Ala Leu Leu Cys Leu Cys Met Leu Leu Cys Gln Glu Ser Pro
210 215 220
Arg Trp Leu Ala Arg Arg Asp Arg Trp Glu Asp Thr Lys Arg Val Leu
225 230 235 240
Ser Arg Ile Arg Asn Leu Pro Pro Asp His Pro Tyr Ile Gln Asp Glu
245 250 255
Phe Gln Glu Ile Val Ala Gln Leu Glu His Glu Arg Arg Leu Ile Gly
260 265 270
Asp Ala Ser Phe Trp Asn Leu Gln Arg Glu Met Trp Thr Ile Ala Gly
275 280 285
Asn Arg Arg Arg Val Leu Ile Ser Ile Ile Leu Met Ile Cys Gln Gln
290 295 300
Met Thr Gly Thr Asn Ala Ile Asn Thr Tyr Ala Pro Thr Ile Phe Lys
305 310 315 320
Asn Leu Gly Leu Thr Gly Thr Ser Thr Ser Leu Phe Ser Thr Gly Val
325 330 335
Tyr Gly Ile Val Lys Val Thr Ser Cys Ile Ile Phe Leu Leu Phe Met
340 345 350
Ala Asp Ser Leu Gly Arg Arg Arg Ser Leu Leu Trp Thr Ser Ile Ala
355 360 365
Gln Gly Leu Ala Met Phe Tyr Ile Gly Leu Tyr Val Arg Ile Ala Pro
370 375 380
Pro Lys Glu Gly Glu Ser Val Pro Pro Ala Gly Tyr Phe Ala Leu Val
385 390 395 400
Cys Ile Phe Leu Phe Ala Ala Phe Phe Gln Phe Gly Trp Gly Pro Ala
405 410 415
Cys Trp Ile Tyr Ala Ser Glu Ile Pro Ala Ala Arg Leu Arg Ser Leu
420 425 430
Asn Val Ala Tyr Ala Ala Ala Thr Gln Trp Leu Phe Asn Phe Val Val
435 440 445
Ala Arg Thr Val Pro Val Met Ile Val Thr Met Gly Glu Gly Gly Tyr
450 455 460
Gly Thr Tyr Leu Leu Phe Gly Ser Phe Cys Phe Ser Met Phe Val Phe
465 470 475 480
Val Trp Phe Phe Val Pro Glu Thr Lys Gly Val Ser Leu Glu Ala Met
485 490 495
Asp Lys Leu Phe Gly Val Thr Asp Glu Ser Ser Lys Ser Leu Thr Val
500 505 510
Asp Glu Asp Ala Lys Glu Lys Glu Lys Asp Gly Pro His Ala Arg Gln
515 520 525
Thr Glu Val Val Ala
530
<210> 23
<211> 512
<212> PRT
<213> Sporotrichum thermophile
<400> 23
Met Lys Lys Phe Leu Gly Leu Arg Gly Gln Ala Leu Asn Leu Ala Val
1 5 10 15
Gly Thr Ile Ala Gly Cys Asp Phe Leu Leu Phe Gly Tyr Asp Gln Gly
20 25 30
Val Met Gly Gly Ile Leu Thr Leu Lys Val Phe Leu Asp Ala Phe Pro
35 40 45
Met Ile Asn Pro Glu Ala Ala Gly Leu Ser His Asp Glu Ser Ser Thr
50 55 60
Arg Ser Thr Tyr Gln Gly Ile Ala Val Ala Ser Tyr Asn Leu Gly Cys
65 70 75 80
Phe Leu Gly Ala Ile Ile Thr Ile Phe Ile Gly Asn Pro Leu Gly Arg
85 90 95
Lys Arg Val Ile Met Leu Gly Thr Ser Val Met Val Ile Gly Ala Ile
100 105 110
Leu Gln Ala Ser Ser Thr Thr Leu Pro Gln Phe Ile Val Gly Arg Ile
115 120 125
Ile Thr Gly Leu Gly Asn Gly Gly Asn Thr Ser Thr Val Pro Thr Trp
130 135 140
Gln Ser Glu Thr Ser Lys Ala His Lys Arg Gly Lys Met Ile Phe Phe
145 150 155 160
Cys Ala Ile Ile Leu Ala Phe Ile Pro Phe Leu Pro Glu Ser Pro Arg
165 170 175
Trp Leu Ile Leu Lys Gly Arg Glu Asp Glu Ala Arg Glu Val Ile Ala
180 185 190
Ala Leu Glu Asp Thr Asp Thr Ser Asp Arg Ile Val Glu Asn Glu Phe
195 200 205
Leu Ala Ile Lys Glu Thr Val Leu Glu Met Ser Lys Gly Thr Phe Arg
210 215 220
Asp Leu Phe Thr Met Asp Lys Asn Arg Asn Leu His Arg Thr Leu Leu
225 230 235 240
Ala Tyr Phe Asn Gln Val Phe Gln Gln Ile Ser Gly Ile Asn Leu Ile
245 250 255
Thr Tyr Tyr Ala Ala Val Ile Tyr Lys Gly Leu Gly Met Ser Asp Phe
260 265 270
Leu Ser Arg Leu Leu Ala Ala Leu Asn Gly Thr Glu Tyr Phe Leu Ala
275 280 285
Ser Trp Pro Ala Val Phe Leu Val Glu Arg Val Gly Arg Arg Asn Leu
290 295 300
Met Leu Phe Gly Ala Val Gly Gln Ala Ala Thr Met Ala Ile Leu Ala
305 310 315 320
Gly Val Asn Ser Arg Gln Glu Thr Gly Phe Gln Ile Ala Gly Ile Val
325 330 335
Phe Leu Phe Val Phe Asn Thr Phe Phe Ala Val Gly Trp Leu Gly Met
340 345 350
Thr Trp Leu Tyr Pro Ala Glu Ile Val Pro Leu Arg Ile Arg Ala Pro
355 360 365
Ala Asn Ala Leu Ser Thr Ser Ala Asn Trp Ile Phe Asn Phe Leu Val
370 375 380
Val Met Ile Thr Pro Val Ala Phe Asn Asn Ile Gly Tyr Gln Thr Tyr
385 390 395 400
Ile Ile Phe Ala Val Ile Asn Ala Phe Met Val Pro Cys Val Tyr Phe
405 410 415
Phe Tyr Pro Glu Thr Ala Tyr Arg Ser Leu Glu Glu Met Asp Asn Ile
420 425 430
Phe His Lys Val Ala Asp Gly Trp Lys Gly Val Phe Thr Val Val His
435 440 445
Gln Ala Lys Val Glu Pro Arg Trp Tyr Gly Lys Asn Gly Glu Leu Leu
450 455 460
Val Asp Tyr Gln Gln Thr Glu Glu His Arg Arg His Leu Gln Gln Gln
465 470 475 480
Glu Gly Ala Val Thr Ala Ser Glu Lys Arg Ser Val Glu Gly Ala Gly
485 490 495
Ser Gly Ser Gly Ser Gly Asp Val Lys Gln Asp Glu Tyr Lys Asp Cys
500 505 510
<210> 24
<211> 519
<212> PRT
<213> Sporotrichum thermophile
<400> 24
Met Glu Ser Thr His Glu Pro Ala Asp Pro Ile Ala Lys Gly Val Leu
1 5 10 15
Ala Thr Ala Lys Gln Ser Trp His Asp Leu Phe Ile Phe Lys Gln Arg
20 25 30
Val Val Val Thr Asn Glu Leu Gly Glu Thr Ser Thr Glu Trp Ala Arg
35 40 45
Pro Val Pro Leu Arg Asn Pro Ile Ser Leu Leu Ala Gln Leu Ser Ala
50 55 60
Arg Asn Trp Leu Phe Phe Ile Val Gly Phe Leu Ala Trp Val Ala Asp
65 70 75 80
Ala Tyr Asp Phe His Ala Leu Ser Ile Gln Gln Val Lys Leu Ala Glu
85 90 95
Phe Tyr Asn Thr Thr Lys Thr Asn Ile Ser Thr Ala Ile Thr Leu Thr
100 105 110
Leu Leu Leu Arg Ser Val Gly Ala Ala Phe Phe Gly Leu Ala Gly Asp
115 120 125
Lys Trp Gly Arg Lys Trp Pro Met Val Ala Asn Met Ile Val Leu Gly
130 135 140
Val Leu Gln Ile Gly Thr Ile Tyr Ser Val Thr Phe Ser Asp Phe Leu
145 150 155 160
Ala Val Arg Ala Leu Phe Gly Leu Phe Met Gly Gly Val Tyr Gly Asn
165 170 175
Ala Ile Ala Met Ala Leu Glu Asn Ser Pro Pro Asp Ala Arg Gly Leu
180 185 190
Met Ser Gly Ile Leu Gln Gln Gly Tyr Ser Leu Gly Tyr Val Ile Ala
195 200 205
Ala Cys Ala Asn Leu Gly Val Gly Gly Gly Asp Asn Ser Trp Lys Thr
210 215 220
Val Phe Trp Ile Gly Ala Gly Leu Ser Ile Gly Val Gly Leu Val Arg
225 230 235 240
Cys Phe Phe Pro Glu Ser Gln Gln Phe Leu Glu Ala Arg Ala Ala Gly
245 250 255
Lys Gly Gln Ala Ser Ala Ser Ala Phe Trp Lys Glu Thr Lys Met Met
260 265 270
Leu Ala Gln Glu Trp Lys Met Cys Val Tyr Cys Ile Ile Leu Met Thr
275 280 285
Trp Phe Asn Tyr Tyr Ser His Thr Ser Gln Asp Ser Tyr Thr Thr Phe
290 295 300
Met Leu Thr Gln Lys Glu Leu Asp Asn Asp Gly Ala Ser Arg Ala Ser
305 310 315 320
Ile Leu Met Lys Val Gly Ala Cys Val Gly Gly Thr Ile Ile Gly Tyr
325 330 335
Ile Ser Gln Trp Phe Gly Arg Arg Arg Thr Ile Ile Val Ala Ala Leu
340 345 350
Ile Ser Gly Leu Ile Ile Pro Ala Trp Ile Leu Pro Glu Gly Glu Arg
355 360 365
Ser Leu Ser Val Thr Gly Phe Phe Met Gln Phe Phe Val Gln Gly Ala
370 375 380
Trp Gly Val Ile Pro Ile His Leu Asn Glu Leu Ser Pro Pro Ala Phe
385 390 395 400
Arg Ser Ser Phe Pro Gly Leu Thr Tyr Gln Leu Gly Asn Met Ile Ser
405 410 415
Ser Pro Ser Ala Gln Ile Val Asn Ala Ile Ala Glu Ser His Ser Val
420 425 430
Thr Ser Lys Ser Gly Lys Ser Val Asn Ala Tyr Gly Pro Thr Met Gly
435 440 445
Ile Ala Thr Ala Ile Ile Ala Thr Gly Ile Ala Val Thr Thr Ala Leu
450 455 460
Gly Pro Glu Lys Arg Gly Arg Glu Phe Glu Lys Thr Leu Pro Ala Gly
465 470 475 480
Met Asn Ile Ile Gln Gly Gly Lys Ala Val Asp Asp Leu Glu Lys Gly
485 490 495
Asp Ser Arg Asp Glu Lys Pro Val Val Gly Glu Val Glu Gly Gly Asn
500 505 510
Asp Gly Ser Gly Glu Lys Lys
515
<210> 25
<211> 488
<212> PRT
<213> Sporotrichum thermophile
<400> 25
Met Ala Asp Glu Lys Arg Met Gly Ser Ser Asp Ser Asp Lys Ala Ala
1 5 10 15
Val Gln His Ser Glu Thr Leu Pro Gly Val Ser Ser Thr Ala Ala Glu
20 25 30
Arg Gly Phe Ala Ala Thr Asp Gln Asn Gly Gln Pro Ile Val Gln Phe
35 40 45
Asp Leu Lys Ala Glu Ala Arg Leu Arg Arg Lys Leu Asp Leu Phe Ile
50 55 60
Val Pro Thr Val Ser Leu Leu Tyr Leu Phe Cys Phe Ile Asp Arg Ala
65 70 75 80
Asn Ile Gly Asn Ala Arg Ile Ala Gly Leu Glu Lys Asp Leu Asn Leu
85 90 95
Thr Gly Tyr Asp Tyr Asn Ala Leu Leu Ser Val Phe Tyr Ile Ser Tyr
100 105 110
Ile Val Phe Glu Ile Pro Ser Asn Ile Ala Cys Lys Trp Ile Gly Pro
115 120 125
Gly Trp Phe Ile Pro Ala Ile Ser Leu Gly Phe Gly Val Val Ser Leu
130 135 140
Ala Thr Ala Phe Val Asp Asn Phe Ala Gln Ala Ala Gly Val Arg Phe
145 150 155 160
Leu Leu Gly Val Phe Glu Ala Gly Met Met Pro Gly Ile Ala Tyr Tyr
165 170 175
Leu Ser Arg Trp Tyr Arg Arg Ala Glu Leu Thr Phe Arg Leu Ser Leu
180 185 190
Tyr Ile Val Met Ala Pro Met Ala Gly Ala Phe Gly Gly Leu Leu Ala
195 200 205
Ser Gly Ile Leu Ser Leu Asp His Val Gly Gly Val Thr Gly Trp Arg
210 215 220
Met Ile Phe Val Val Glu Gly Ile Ile Thr Ile Gly Leu Ser Val Ile
225 230 235 240
Ser Phe Ile Thr Leu Thr Asp Arg Pro Glu Thr Ala Arg Trp Leu Thr
245 250 255
Gln Glu Glu Lys Asp Leu Ala Ile Ala Arg Val Lys Ser Glu Arg Val
260 265 270
Ala Thr Thr Glu Val Leu Asp Arg Met Asp Thr Lys Lys Leu Ile Gln
275 280 285
Gly Ile Leu Ser Pro Val Thr Leu Ala Thr Ser Phe Met Phe Leu Leu
290 295 300
Asn Asn Ile Thr Gln Leu Phe Thr Val Pro Pro Tyr Val Val Gly Gly
305 310 315 320
Phe Phe Thr Leu Ala Leu Pro Leu Leu Ser Trp Tyr Leu Asp Arg Arg
325 330 335
Gln Ile Ile Ile Leu Leu Ser Thr Pro Leu Val Ile Val Gly Tyr Ser
340 345 350
Met Phe Leu Gly Thr Thr Asn Pro Ser Ala Arg Tyr Gly Ala Thr Phe
355 360 365
Leu Leu Ser Ser Ser Leu Phe Ala Val Gly Ala Leu Ser Asn Ser Gln
370 375 380
Val Ser Ala Asn Val Val Ser Asp Thr Ala Arg Ser Ser Ala Ile Gly
385 390 395 400
Leu Asn Val Met Met Gly Asn Val Gly Gly Leu Ile Ala Thr Trp Ser
405 410 415
Tyr Leu Pro Trp Asp Gly Pro Asn Tyr Lys Ile Gly Asn Gly Leu Asn
420 425 430
Leu Ala Ala Cys Cys Thr Val Leu Ile Leu Ser Ala Val Thr Leu Leu
435 440 445
Trp Met Lys Trp Asp Asn Arg Arg Arg Glu Gly Arg Asn Ala Glu Glu
450 455 460
Glu Leu Ala Gly Met Ser Arg Gln Glu Ile Gln Asp Leu Asp Trp Lys
465 470 475 480
His Pro Ala Phe Arg Trp Arg Pro
485
<210> 26
<211> 546
<212> PRT
<213> Sporotrichum thermophile
<400> 26
Met Pro Lys Ala Arg Ser Arg Val Pro Val Lys Val Asn Val Gly Thr
1 5 10 15
Ser Ala Asp Pro Ile Val Thr Arg Leu Val Gln Glu Asp Lys Ile Pro
20 25 30
Trp Tyr Lys Lys Pro Asn Leu Arg Arg Met Tyr Ile Phe Leu Phe Leu
35 40 45
Cys Cys Met Gly Val Glu Met Thr Ser Gly Phe Asp Ser Gln Leu Ile
50 55 60
Asn Thr Leu Gln Tyr Ala Glu Thr Phe His Lys Tyr Leu Gly Asn Gly
65 70 75 80
Arg Lys Asp Glu Asp Gly Asn Tyr Ala Ile Glu Pro Gly Leu Leu Gly
85 90 95
Phe Val Asn Ser Cys Tyr Gln Leu Gly Ser Ile Phe Ala Val Pro Ile
100 105 110
Ala Pro Trp Phe Ala Gln Arg Phe Gly Arg Arg Trp Ser Ile Met Leu
115 120 125
Gly Ser Leu Ile Met Val Gly Gly Ala Ile Ile Gln Gly Phe Ala Gln
130 135 140
His Val Ala Met Tyr Ile Ile Ala Arg Met Ile Leu Gly Met Gly Ile
145 150 155 160
Leu Phe Cys Ile Ile Ser Gly Ala Ala Leu Ile Gly Glu Leu Gly His
165 170 175
Pro Lys Glu Arg Ala Val Leu Thr Ser Leu Phe Asn Ser Ser Tyr Phe
180 185 190
Ile Gly Gln Ile Leu Ala Ser Ala Ile Thr Ile Gly Thr Thr Glu Met
195 200 205
Lys Thr Asn Trp Ala Trp Arg Leu Pro Ser Leu Leu Gln Ile Cys Pro
210 215 220
Ser Leu Leu Gln Ile Val Thr Val Phe Phe Leu Pro Glu Ser Pro Arg
225 230 235 240
Phe Leu Ile Ser Lys Asp Arg Asp Asp Asp Ala Lys Glu Val Leu Ile
245 250 255
Lys Tyr His Ala Glu Gly Asp Ala Ser Ser Leu Leu Val Gln Ala Glu
260 265 270
Ile Val Gln Ile Arg Glu Thr Ile Arg Thr Glu Met Glu Val Ser Asn
275 280 285
Gln Ser Trp Met Glu Leu Val Ser Thr Tyr Gly Met Arg Arg Arg Leu
290 295 300
Val Ile Thr Leu Phe Ile Gly Leu Phe Thr Gln Leu Ser Gly Asn Thr
305 310 315 320
Leu Leu Ser Tyr Tyr Ser Gly Lys Leu Phe Glu Met Met Gly Tyr Thr
325 330 335
Glu Ala Ser Val Lys Thr Arg Ile Asn Val Ala Asn Ala Cys Trp Ser
340 345 350
Leu Leu Asn Ala Thr Thr Ile Ala Phe Leu Val Pro Tyr Phe Lys Arg
355 360 365
Arg His Met Phe Met Thr Ser Ala Leu Ser Met Cys Ala Val Phe Ile
370 375 380
Ala Ile Thr Val Ser Leu Glu Arg Thr Gln Ala Ala Gln Asp Ala Gly
385 390 395 400
Phe Lys Asn Thr Ala Ala Gly Ile Ser Gly Leu Phe Trp Tyr Phe Ala
405 410 415
Phe Ala Pro Cys Tyr Asn Met Gly Asn Asn Ala Leu Thr Tyr Thr Tyr
420 425 430
Leu Val Glu Leu Trp Pro Tyr Ser His Arg Ser Arg Gly Ile Gly Val
435 440 445
Gln Gln Ile Phe Gly Lys Leu Gly Gly Phe Phe Ser Thr Asn Val Asn
450 455 460
Ser Ile Ala Leu Asp Ala Ile Arg Trp Lys Tyr Met Ala Ile Tyr Cys
465 470 475 480
Gly Trp Ile Phe Phe Glu Phe Leu Ile Val Phe Phe Leu Tyr Pro Glu
485 490 495
Thr Ser Gly Arg Thr Leu Glu Glu Leu Ala Phe Leu Phe Glu Asp Ala
500 505 510
Ser Leu Asn Glu Lys Ala Ala Ala Ala Val Glu Lys Gln Ile His Tyr
515 520 525
Gly Asp Glu Lys Val Val His Glu Glu Gly Gln Pro Ala Ala Lys Ser
530 535 540
Val Val
545
<210> 27
<211> 481
<212> PRT
<213> Sporotrichum thermophile
<400> 27
Met Leu Ser Ser Gly Phe Trp Lys Arg Arg Ser Leu Arg Val Pro Asp
1 5 10 15
Asn Gln Arg Thr Lys Ala Ala Glu Leu Thr Leu Arg Glu Ser Leu Tyr
20 25 30
Pro Leu Ser Leu Val Thr Ile Leu Phe Phe Leu Trp Gly Phe Ser Tyr
35 40 45
Gly Leu Leu Asp Thr Leu Asn Lys His Phe Gln Asn Thr Leu Gly Ile
50 55 60
Thr Lys Thr Arg Ser Ser Gly Leu Gln Ala Ala Tyr Phe Gly Ala Tyr
65 70 75 80
Pro Leu Ala Ser Leu Gly His Ala Ala Trp Ile Leu Arg His Tyr Gly
85 90 95
Tyr Arg Ala Val Phe Ile Trp Gly Leu Phe Leu Tyr Gly Leu Gly Ala
100 105 110
Leu Leu Ala Ile Pro Ser Ile Met His His Ser Phe Ala Gly Phe Cys
115 120 125
Val Cys Ile Phe Ile Ile Gly Asn Gly Leu Gly Ser Leu Glu Thr Ala
130 135 140
Ala Asn Pro Tyr Ile Thr Val Cys Gly Pro Pro Lys Phe Ser Glu Ile
145 150 155 160
Arg Ile Asn Val Ala Gln Ala Phe Asn Gly Ile Gly Thr Val Val Ala
165 170 175
Pro Val Leu Gly Ser Tyr Val Phe Phe Thr Phe Asp Asp Gln Thr Ala
180 185 190
Leu Arg Asn Val Gln Trp Val Tyr Leu Ala Ile Ala Cys Phe Val Phe
195 200 205
Leu Leu Ala Gly Val Phe Phe Leu Ser Val Ile Pro Glu Ile Thr Asp
210 215 220
Ala Asp Met Ala Phe Gln Ala Ala Glu Thr His Ala Gly Ala Asp Asp
225 230 235 240
Arg Pro Phe His Thr Gln Tyr Arg Leu Phe His Ala Ala Phe Ala Gln
245 250 255
Phe Cys Tyr Thr Gly Ala Gln Val Ala Ile Ala Gly Tyr Phe Ile Asn
260 265 270
Tyr Ala Thr Glu Thr Arg Pro Asn Thr Asp Ser Ser Leu Gly Ser Lys
275 280 285
Phe Leu Ala Gly Ser Gln Ala Gly Phe Ala Val Gly Arg Phe Gly Gly
290 295 300
Ala Ala Met Met Gln Phe Ile Lys Pro Arg Lys Val Phe Ala Leu Phe
305 310 315 320
Met Thr Met Cys Ile Val Phe Ser Ala Pro Ala Ile Thr Gln Arg Gly
325 330 335
Asn Ala Gly Leu Ser Met Leu Tyr Leu Val Met Phe Phe Glu Ser Ile
340 345 350
Cys Phe Pro Thr Ile Ile Ala Leu Gly Met Arg Gly Leu Gly Arg His
355 360 365
Thr Lys Arg Gly Ser Gly Trp Ile Val Ala Gly Val Leu Gly Gly Ala
370 375 380
Cys Val Pro Pro Leu Met Gly Ala Ala Ala Asp Ala Arg Gly Thr Gly
385 390 395 400
Phe Ser Met Leu Val Pro Leu Cys Phe Phe Val Ala Ala Trp Thr Tyr
405 410 415
Ala Leu Ala Val Asn Phe Ala Pro Pro Tyr Arg Ser Val Val Asp Ala
420 425 430
Phe Ser Thr Thr Asp Val Gly Leu Arg Glu Lys Gln Arg Glu Asp Val
435 440 445
Gly Ala Glu Lys Gly Gly Glu Ala Gly Gly Lys Gly Gly Val Thr Gly
450 455 460
Pro Glu Asp Ala Ser Glu Asp Lys Pro Asp Val Val Asn Ser Glu Lys
465 470 475 480
Val
<210> 28
<211> 419
<212> PRT
<213> Sporotrichum thermophile
<400> 28
Met Leu Ser Ser Leu Arg Ile Ala Ser Arg Arg Ala Ala Val Ala Arg
1 5 10 15
Asn Phe Ser Ala Val Arg Ala Ala Ser Thr Trp Ala Asn Val Pro Gln
20 25 30
Gly Pro Pro Val Cys Ile Thr Glu Ala Phe Lys Ala Asp Pro Phe Glu
35 40 45
Lys Lys Ile Asn Leu Gly Val Gly Ala Tyr Arg Asp Asp Lys Gly Lys
50 55 60
Pro Tyr Val Leu Pro Ser Val Arg Lys Ala Glu Glu Lys Val Ile Ala
65 70 75 80
Ser Arg Leu Asn Lys Glu Tyr Ala Gly Ile Thr Gly Val Pro Glu Phe
85 90 95
Thr Lys Ala Ala Ala Val Leu Ala Tyr Gly Lys Asp Ser Ser Ala Leu
100 105 110
Asp Arg Leu Ala Ile Thr Gln Ser Ile Ser Gly Thr Gly Ala Leu Arg
115 120 125
Ile Gly Ala Ala Phe Leu Ser Arg Phe Tyr Pro Gly Ala Lys Thr Ile
130 135 140
Tyr Ile Pro Thr Pro Ser Trp Ala Asn His Ala Ala Val Phe Lys Asp
145 150 155 160
Ser Gly Leu Gln Val Glu Lys Tyr Ala Tyr Tyr Asn Lys Asp Thr Ile
165 170 175
Arg Leu Asp Phe Glu Gly Met Ile Ala Asp Ile Asn Lys Ala Pro Asn
180 185 190
Gly Ser Ile Phe Leu Phe His Ala Cys Ala His Asn Pro Thr Gly Val
195 200 205
Asp Pro Thr Gln Glu Gln Trp Lys Glu Ile Glu Ala Ala Val Lys Ala
210 215 220
Lys Gly His Phe Ala Phe Phe Asp Met Ala Tyr Gln Gly Phe Ala Ser
225 230 235 240
Gly Asp Ile His Arg Asp Ala Phe Ala Val Arg Tyr Phe Val Glu Lys
245 250 255
Gly His Asn Ile Cys Leu Ala Gln Ser Phe Ala Lys Asn Met Gly Leu
260 265 270
Tyr Gly Glu Arg Thr Gly Ala Phe Ser Ile Val Cys Ala Asp Ala Glu
275 280 285
Glu Arg Lys Arg Val Asp Ser Gln Ile Lys Ile Leu Val Arg Pro Met
290 295 300
Tyr Ser Asn Pro Pro Ile His Gly Ala Arg Ile Ala Ala Glu Ile Leu
305 310 315 320
Asn Thr Pro Glu Leu Tyr Asp Gln Trp Leu Val Glu Val Lys Glu Met
325 330 335
Ala Asn Arg Ile Ile Thr Met Arg Ala Leu Leu Lys Glu Asn Leu Glu
340 345 350
Lys Leu Gly Ser Lys His Asp Trp Ser His Ile Thr Ser Gln Ile Gly
355 360 365
Met Phe Ala Tyr Thr Gly Leu Thr Pro Glu Gln Met Glu Lys Leu Ala
370 375 380
Lys Glu His Ser Val Tyr Ala Thr Arg Asp Gly Arg Ile Ser Val Ala
385 390 395 400
Gly Ile Thr Thr Asp Asn Val Gly Arg Leu Ala Glu Ala Ile Phe Lys
405 410 415
Val Lys Gly
<210> 29
<211> 522
<212> PRT
<213> Sporotrichum thermophile
<400> 29
Met Gly Ile Phe Ala Phe Asn Lys Gln Lys Pro Asn Ala Glu Ala Thr
1 5 10 15
Ala Val Ala Gln Glu Glu Ala Pro Gln Phe Glu Arg Val Asp Trp Lys
20 25 30
Arg Asp Pro Gly Leu Arg Lys Leu Tyr Phe Tyr Ala Phe Val Leu Cys
35 40 45
Ile Ala Ser Ala Thr Thr Gly Tyr Asp Gly Met Phe Phe Asn Ser Val
50 55 60
Gln Asn Phe Glu Thr Trp Glu Asn Tyr Phe Asn His Pro Thr Gly Ser
65 70 75 80
Lys Leu Gly Val Leu Gly Ala Leu Tyr Gln Ile Gly Ser Leu Ala Ser
85 90 95
Ile Pro Leu Val Pro Ile Ile Ala Asp Arg Val Gly Arg Lys Ile Pro
100 105 110
Ile Ala Ile Gly Cys Val Ile Met Ile Val Gly Ala Val Leu Gln Ala
115 120 125
Ala Cys Arg Asn Leu Gly Thr Phe Met Gly Gly Arg Phe Leu Leu Gly
130 135 140
Phe Gly Asn Ser Leu Ala Gln Leu Cys Ser Pro Met Leu Leu Thr Glu
145 150 155 160
Leu Ala His Pro Gln His Arg Gly Arg Leu Thr Thr Val Tyr Asn Cys
165 170 175
Leu Trp Asn Val Gly Ala Leu Val Val Ala Trp Val Ser Phe Gly Thr
180 185 190
Asp Tyr Leu Lys Ser Asp Trp Ser Trp Arg Ile Pro Ala Leu Ile Gln
195 200 205
Ala Phe Pro Ser Val Ile Gln Leu Leu Phe Ile Phe Trp Val Pro Glu
210 215 220
Ser Pro Arg Tyr Leu Met Ala Lys Asp Lys His Glu Arg Ala Leu Ala
225 230 235 240
Ile Leu Ala Lys Tyr His Ala Asn Gly Asp Ala Asn His Pro Thr Val
245 250 255
Gln Phe Glu Tyr Arg Glu Ile Lys Glu Thr Leu Arg Leu Glu Phe Glu
260 265 270
Ala Ser Lys Ser Ser Ser Tyr Leu Asp Phe Val Arg Thr Arg Gly Asn
275 280 285
Arg Tyr Arg Leu Ala Val Leu Ile Ser Leu Gly Ile Phe Ser Gln Trp
290 295 300
Ser Gly Asn Ala Ile Ile Ser Asn Tyr Ser Ser Lys Leu Tyr Asp Thr
305 310 315 320
Ala Gly Val Thr Gly Ser Thr Gln Lys Leu Gly Leu Ser Ala Gly Gln
325 330 335
Thr Gly Leu Ser Leu Ile Ile Ser Val Thr Met Ala Leu Leu Val Asp
340 345 350
Lys Phe Gly Arg Arg Pro Met Phe Leu Thr Ser Thr Ala Gly Met Phe
355 360 365
Cys Thr Phe Ile Phe Trp Thr Leu Thr Ser Gly Leu Tyr Glu Glu His
370 375 380
Asn Ala Asp Gly Ala Arg Tyr Ala Met Ile Leu Phe Ile Trp Ile His
385 390 395 400
Gly Ile Phe Tyr Ser Ile Ser Trp Ser Gly Leu Leu Val Gly Tyr Ala
405 410 415
Ile Glu Val Leu Pro Tyr Lys Leu Arg Ala Lys Gly Leu Met Ile Met
420 425 430
Asn Leu Thr Val Gln Ala Ala Leu Thr Leu Asn Thr Tyr Ala Asn Pro
435 440 445
Val Ala Phe Asp Ala Phe Glu Gly His Ser Trp Lys Leu Tyr Ile Ile
450 455 460
Tyr Thr Ile Trp Ile Phe Leu Glu Leu Cys Phe Val Trp Lys Met Tyr
465 470 475 480
Ile Glu Thr Lys Gly Pro Thr Leu Glu Glu Leu Ala Lys Ile Ile Asp
485 490 495
Gly Asp Glu Ala Ala Val Ala His Val Asp Ile Lys Gln Val Glu Lys
500 505 510
Glu Thr His Ile Asn Glu Glu Lys Ser Val
515 520
<210> 30
<211> 554
<212> PRT
<213> Sporotrichum thermophile
<400> 30
Met Ser Ser Ser Glu Lys Glu Ala Thr Gly Pro Val Ala Ala His Val
1 5 10 15
Gly Asn Leu Ala Thr Thr Gln Asp Val Glu Lys Ile Glu Ala Pro Val
20 25 30
Thr Trp Lys Ala Tyr Leu Ile Cys Ala Phe Ala Ser Phe Gly Gly Ile
35 40 45
Phe Phe Gly Tyr Asp Ser Gly Tyr Ile Asn Gly Val Leu Ala Ser Lys
50 55 60
Leu Phe Ile Asn Ala Val Glu Gly Ala Gly Lys Asp Ala Ile Ser Glu
65 70 75 80
Ser His Ser Ser Leu Ile Val Ser Ile Leu Ser Cys Gly Thr Phe Phe
85 90 95
Gly Ala Leu Ile Ala Gly Asp Leu Ala Asp Phe Ile Gly Arg Lys Tyr
100 105 110
Thr Val Ile Leu Gly Cys Leu Ile Tyr Ile Ile Gly Cys Val Ile Gln
115 120 125
Ile Ile Thr Gly Leu Gly Asn Ala Leu Gly Ala Ile Val Ala Gly Arg
130 135 140
Leu Ile Ala Gly Ile Gly Val Gly Phe Glu Ser Ala Ile Val Ile Leu
145 150 155 160
Tyr Met Ser Glu Ile Cys Pro Arg Lys Val Arg Gly Ala Leu Val Ala
165 170 175
Gly Tyr Gln Phe Cys Ile Thr Ile Gly Leu Met Leu Ala Ser Cys Val
180 185 190
Val Tyr Gly Thr Gln Asn Arg Gln Asp Thr Gly Gln Tyr Arg Ile Pro
195 200 205
Ile Gly Ile Gln Phe Ile Trp Ala Leu Ile Leu Gly Gly Gly Leu Leu
210 215 220
Cys Leu Pro Asp Ser Pro Arg Tyr Phe Val Lys Arg Gly Arg Leu Ala
225 230 235 240
Asp Ala Thr Ser Ala Leu Ser Arg Leu Arg Gly Gln Pro Glu Asp Ser
245 250 255
Glu Tyr Ile Gln Val Glu Leu Ala Glu Ile Val Ala Asn Glu Glu Tyr
260 265 270
Glu Arg Gln Leu Ile Pro Ser Thr Thr Trp Phe Gly Ser Trp Ala Asn
275 280 285
Cys Phe Lys Gly Ser Leu Phe Lys Ala Asn Ser Asn Leu Arg Lys Thr
290 295 300
Ile Leu Gly Thr Ser Leu Gln Met Met Gln Gln Trp Thr Gly Val Asn
305 310 315 320
Phe Ile Phe Tyr Tyr Ser Thr Pro Phe Leu Lys Ser Thr Gly Ala Ile
325 330 335
Asp Asp Pro Phe Leu Met Ser Met Val Phe Thr Ile Ile Asn Val Phe
340 345 350
Ser Thr Pro Ile Ser Phe Tyr Thr Val Glu Arg Phe Gly Arg Arg Thr
355 360 365
Ile Leu Phe Trp Gly Ala Leu Gly Met Leu Ile Cys Gln Phe Leu Val
370 375 380
Ala Ile Val Gly Val Thr Val Gly Phe Asn His Thr His Pro Ala Pro
385 390 395 400
Thr Ala Asp Asp Pro Glu Ala Thr Leu Ala Asn Asn Ile Ser Ala Val
405 410 415
Asn Ala Gln Ile Ala Phe Ile Ala Ile Phe Ile Phe Phe Phe Ala Ser
420 425 430
Thr Trp Gly Pro Gly Ala Trp Ile Val Ile Gly Glu Ile Phe Pro Leu
435 440 445
Pro Ile Arg Ser Arg Gly Val Gly Leu Ser Thr Ala Ser Asn Trp Leu
450 455 460
Trp Asn Thr Ile Ile Ala Val Ile Thr Pro Tyr Met Val Gly Glu Asp
465 470 475 480
Arg Gly Asn Met Lys Ser Ser Val Phe Phe Val Trp Gly Gly Leu Cys
485 490 495
Thr Cys Ala Phe Val Tyr Thr Tyr Phe Leu Val Pro Glu Thr Lys Gly
500 505 510
Leu Ser Leu Glu Gln Val Asp Lys Met Met Glu Glu Thr Thr Pro Arg
515 520 525
Thr Ser Ala Lys Trp Lys Pro Thr Thr Thr Phe Ala Ala Ser His Pro
530 535 540
Thr Asp Leu Lys Gln Gly Glu Ala Ala Val
545 550
<210> 31
<211> 537
<212> PRT
<213> Sporotrichum thermophile
<400> 31
Met Gly Thr Ser Arg Asp Glu Lys Glu Thr Val Val Ala Asp His Ala
1 5 10 15
Asp Asp Asp Ala Leu Arg Glu Ala Asp Leu Ala Val Gln Val Ala His
20 25 30
Asp Ala Asp Gly Thr Val Tyr Ser Pro Trp Ser Leu Arg Met Ile Arg
35 40 45
Leu Tyr Leu Val Leu Ser Leu Ser Tyr Leu Cys Gly Cys Leu Asn Gly
50 55 60
Tyr Asp Gly Ser Leu Met Gly Gly Leu Asn Gly Met Thr Ser Tyr Gln
65 70 75 80
Arg Tyr Phe His Met Ser Thr Ala Gly Ser Thr Thr Gly Leu Ile Phe
85 90 95
Ala Met Tyr Asn Ile Gly Ser Val Ala Ala Val Phe Phe Thr Gly Pro
100 105 110
Val Asn Asp Tyr Phe Gly Arg Arg Trp Gly Met Phe Val Gly Ala Leu
115 120 125
Leu Val Ile Val Gly Thr Cys Val Gln Ala Pro Cys Thr Thr Arg Gly
130 135 140
Gln Phe Leu Ala Gly Arg Phe Val Leu Gly Phe Gly Val Ser Phe Cys
145 150 155 160
Cys Val Ser Ala Pro Cys Tyr Val Ser Glu Met Ala His Pro Lys Trp
165 170 175
Arg Gly Thr Leu Thr Gly Leu Tyr Asn Cys Thr Trp Tyr Ile Gly Ser
180 185 190
Ile Val Ala Ser Trp Val Val Tyr Gly Cys Ser Tyr Ile Asp Thr Leu
195 200 205
Asp Ala Trp Arg Ile Pro Ile Trp Cys Gln Met Val Thr Ser Gly Leu
210 215 220
Val Cys Leu Gly Val Phe Trp Leu Pro Glu Ser Pro Arg Trp Leu Met
225 230 235 240
Ala Gln Asp Arg His Asp Asp Ala Ala Arg Val Leu Ala Thr Tyr His
245 250 255
Gly Glu Gly Arg Ala Asp His Pro Leu Val Lys Leu Gln Met Gln Glu
260 265 270
Met Met Asn Gln Ile Ser Thr Glu Ala Ser Asp Lys Lys Trp Tyr Asp
275 280 285
Tyr His Glu Leu Trp Asn Thr His Ser Ala Arg Arg Arg Leu Ile Cys
290 295 300
Val Ile Gly Met Ala Val Phe Gly Gln Ile Ser Gly Asn Ser Leu Ser
305 310 315 320
Ser Tyr Tyr Leu Val Asn Met Leu Lys Ser Ala Gly Ile Thr Glu Glu
325 330 335
Arg Arg Val Leu Ala Leu Asn Gly Val Asn Pro Ala Leu Ser Phe Leu
340 345 350
Gly Ala Ile Leu Gly Ala Arg Met Thr Asp Val Val Gly Arg Arg Pro
355 360 365
Leu Leu Leu Tyr Thr Ile Val Phe Ala Ser Val Cys Phe Ala Val Ile
370 375 380
Thr Gly Thr Ser Lys Met Ala Thr Asp Asp Pro Thr Arg Thr Ala Ala
385 390 395 400
Ala Asn Ala Thr Ile Ala Phe Ile Phe Ile Phe Gly Ile Val Phe Ser
405 410 415
Phe Gly Trp Thr Pro Leu Gln Ser Met Tyr Ile Ala Glu Thr Leu Pro
420 425 430
Thr Ala Thr Arg Ala Lys Gly Thr Ala Val Gly Asn Phe Ser Ser Ser
435 440 445
Val Ala Ser Thr Ile Leu Gln Tyr Ala Ser Gly Pro Ala Phe Glu Gly
450 455 460
Ile Gly Tyr Tyr Phe Tyr Leu Val Phe Val Phe Trp Asp Leu Ile Glu
465 470 475 480
Gly Ala Ile Met Tyr Phe Tyr Phe Pro Glu Thr Lys Asp Arg Thr Leu
485 490 495
Glu Glu Leu Glu Glu Val Phe Ser Ala Pro Asn Pro Val Lys Lys Ser
500 505 510
Leu Glu Lys Arg Ser Ala Gln Thr Val Leu Asn Thr Val Gly Ala Ala
515 520 525
Gln Asn Glu Lys Leu Ala Arg Asp Val
530 535
<210> 32
<211> 566
<212> PRT
<213> Sporotrichum thermophile
<400> 32
Met Ala Val Phe Ala Met Gly Trp Gln Lys Pro Asp Asn Val Ala Gly
1 5 10 15
Ser Ser Ala Pro Ala Ile Met Val Gly Leu Phe Val Ala Thr Gly Gly
20 25 30
Leu Leu Phe Gly Tyr Asp Thr Gly Ala Ile Asn Gly Ile Leu Ala Met
35 40 45
Asp Thr Phe Lys Glu Asp Phe Thr Thr Gly Tyr Thr Asp Lys Gln Gly
50 55 60
Lys Pro Gly Leu Tyr Ala Ser Glu Val Ser Leu Ile Val Ala Met Leu
65 70 75 80
Ser Ala Gly Thr Ala Thr Gly Ala Leu Leu Ser Ala Pro Met Gly Asp
85 90 95
Arg Trp Gly Arg Arg Leu Ser Leu Ile Val Ala Ile Gly Val Phe Cys
100 105 110
Val Gly Ala Ile Ile Gln Val Cys Ala Thr Asn Val Ala Met Leu Val
115 120 125
Val Gly Arg Thr Leu Ala Gly Ile Gly Val Gly Val Val Ser Val Leu
130 135 140
Val Pro Leu Tyr Gln Ser Glu Met Ala Pro Lys Trp Ile Arg Gly Thr
145 150 155 160
Leu Val Cys Ala Tyr Gln Leu Ser Ile Thr Ala Gly Leu Leu Ala Ala
165 170 175
Ala Thr Val Asn Ile Leu Thr Tyr Lys Leu Lys Ser Ala Ala Ala Tyr
180 185 190
Arg Ile Pro Ile Gly Leu Gln Leu Thr Trp Ala Leu Val Leu Ala Leu
195 200 205
Gly Leu Val Ile Leu Pro Glu Thr Pro Arg Tyr Leu Val Lys Arg Gly
210 215 220
Leu Lys Glu Ala Ala Ala Leu Ser Leu Ser Arg Leu Arg Arg Leu Asp
225 230 235 240
Ile Thr His Pro Ala Leu Ile Glu Glu Leu Ala Glu Ile Glu Ala Asn
245 250 255
His Glu Tyr Glu Met Ala Leu Gly Pro Asp Thr Tyr Lys Asp Ile Ile
260 265 270
Phe Gly Glu Pro His Leu Gly Arg Arg Thr Leu Thr Gly Cys Gly Leu
275 280 285
Gln Met Leu Gln Gln Leu Thr Gly Val Asn Phe Ile Met Tyr Tyr Gly
290 295 300
Thr Thr Phe Phe Tyr Gly Ala Gly Ile Gly Asn Ala Phe Thr Val Ser
305 310 315 320
Leu Ile Met Gln Val Ile Asn Leu Val Ser Thr Phe Pro Gly Leu Phe
325 330 335
Val Val Glu Ser Trp Gly Arg Arg Lys Leu Leu Ile Val Gly Ser Val
340 345 350
Gly Met Ala Ile Cys Gln Leu Leu Ile Ala Ser Phe Ala Thr Ala Ser
355 360 365
Gly Asn Asp Asn Lys Pro Thr Gln Asn Gln Ile Leu Ile Ile Phe Val
370 375 380
Ala Ile Tyr Ile Phe Phe Phe Ala Ala Ser Trp Gly Pro Val Val Trp
385 390 395 400
Val Val Thr Ser Glu Ile Tyr Pro Leu Lys Val Arg Ala Lys Ser Met
405 410 415
Ser Ile Ser Thr Ala Ser Asn Trp Val Leu Asn Phe Gly Ile Ala Tyr
420 425 430
Gly Thr Pro Tyr Leu Val Asp Thr Ser Asp Gly Ser Pro Asp Leu Gly
435 440 445
Ser Arg Val Phe Phe Val Trp Gly Ala Phe Cys Ile Leu Ser Ile Ala
450 455 460
Phe Val Trp Tyr Met Val Tyr Glu Thr Ser Lys Ile Ser Leu Glu Gln
465 470 475 480
Ile Asp Glu Met Tyr Glu Arg Val Ala His Ala Trp Asn Ser Arg Ser
485 490 495
Phe Glu Pro Ser Trp Ser Phe Gln Gln Met Arg Asp Phe Gly Phe Ser
500 505 510
Asp Ser Gly Ile Pro Pro Ala Glu Pro Gln Leu Glu Leu Gln Gln Ser
515 520 525
Asn Ala Ser Thr Ser Gln Ser Asp Thr Gly Gly Ser Ser Ala Thr His
530 535 540
Ala Thr Ala Ala Asn Pro Gln Asp Ala Lys Met Val Ser Gln Leu Ala
545 550 555 560
Asn Ile Asp Leu Ser Tyr
565
<210> 33
<211> 1404
<212> DNA
<213> Pichia stipitis
<400> 33
atgaagtatt ttcaaatctg gaaatcaggc aaacaagtaa gctacgctgt tacattcact 60
tgtgaattgg catttattct ttttggtatt gaacagggta ttattggtaa tcttattaac 120
aaccaggact tcctaaacac ttttggaaac cccaccggta gttatttagg tattatcgtt 180
tctatctata ccttagggtg tttttttggt tgtgttatga acttcttcat tggtgatcga 240
atgggcagaa gaagcaaaat tgcttcctca atgacagtta tcacaattgg tgttgctctt 300
caatgtagtt ccttttcagt tgaacaattg atgattggaa gatttatcac tgggcttgga 360
actggttggg aaacttctac ttgtccaatg tatcaggcag aactttcacc tccaaaagtt 420
agaggacgtt tggtgtgctc agaagcattg tttgttggag ttggtttaat ctatgcatat 480
tggtttgatt atgctctttc tttcacttct ggtcctattg catggagact tcctcttgcc 540
tctcagattg tgttcgcctt tgttgttttc tgtttcactt tcacaatacc cgaatcccct 600
agatacatgt tttacaaagg agagaaagaa gaagccaaaa gaattttatc ttatgtcttt 660
ggaaagccag gagatcatcc tgacattctt aaggaatgga atgatattaa tgatgctgtt 720
attttggaaa cttcagaagg agctttctcg tgggcaaaac ttttcaagcc cgataaggca 780
agaactggat acagagtctt cttggcatac atgagcatgt ttgcgcaaca gttgagtggt 840
gttaatgtag ttaattacta tattacattt gttttgatta acagtgttgg catcgaagac 900
aacttggccc taattcttgg tggtgttgcc gtcatctgtt tcactgttgg ttcattagtt 960
cctactttct ttgctgatag gatgggaaga agattgcctt cagcagttgg agcttttggc 1020
tgtggtgttt gtatgatgct aatttcaatc ttattaagtt ttcaagacaa tccaaagttg 1080
aagaagagca gtggagctgg tgctgtggct ttctttttcg ttttccaact tgtcttcggc 1140
tccactggta attgtattcc atggctgatg atttcagagc ttatccccct tcatgcacgt 1200
gctaaaggat cttcattagc tacatcaagt aactggcttt ggaatttctt tgttgttgag 1260
atcactccaa ctatcattga aaagttgaag tggaaagcat atttgatctt tatgtgctgc 1320
aacttctcct tcgtaccaat gttttacttt ttctttcccg agacaaagaa ccttacttta 1380
gaagccattg acgatttgtt ctca 1404
<210> 34
<211> 1653
<212> DNA
<213> Pichia stipitis
<400> 34
atgtcctcac aagatttacc ctcgggtgct caaaccccaa tcgatggttc ttccatcctc 60
gaagataaag ttgagcaaag ttcgtcctca aatagccaaa gtgatttagc ttccattcca 120
gcaacaggta tcaaagccta tctcttggtt tgtttcttct gcatgttggt tgcctttggt 180
ggcttcgtat tcggtttcga taccggtaca atttccggtt tccttaatat gtctgatttc 240
ctttccagat ttggtcaaga tggttctgaa ggaaaatatt tgtctgatat cagagtcggt 300
ttgattgttt ccatttttaa cattggttgt gcaattggtg gtattttcct ttctaagata 360
ggagatgttt acggtagaag aattggtatc atttcagcta tggttgtcta cgtcgtcggt 420
attatcatcc agatctcgtc ccaagacaag tggtaccaac ttacaattgg acgtggagtt 480
acaggattag ctgttggtac tgtttcagtg ttgtctccaa tgttcattag tgaaagtgct 540
ccaaagcatt tgagaggtac tttggtatac tgttaccaat tatgtatcac cttaggtatt 600
ttcattggtt actgtgtcac ttatggaacc aaagatttaa atgattcaag acaatggaga 660
gttcctttgg gcttatgctt cctttgggct attttcttag ttgtcggtat gttggctatg 720
ccagaatccc caagattctt aattgaaaag aagagaatcg aagaagccaa gaagtccctt 780
gcaagatcca acaagttatc tccggaagat ccaggtgtct acactgaact tcaattgatt 840
caggctggta ttgacagaga agctgctgca ggttctgctt cgtggatgga attgatcact 900
ggtaagccag ctattttcag aagagttatc atgggaatta tcttgcagtc tttgcaacaa 960
ttaactggtg tcaactattt cttctattac ggaactacaa tcttccaagc tgttggtttg 1020
caagattcct tccagacttc catcatctta ggtacagtca actttctttc tacatttgtt 1080
ggtatttggg ccattgaaag atttggaaga agacaatgtt tgttagtcgg ttctgctggt 1140
atgttcgttt gtttcatcat ttactccgtt attggtacaa ctcatttgtt cattgatgga 1200
gtagtagata acgacaacac ccgtcaactg tctggtaatg ctatgatctt tatcacttgt 1260
ttgttcatct tcttctttgc ctgtacatgg gctggaggtg tttttaccat catttccgaa 1320
tcatatccat tgagaatcag atccaaggca atgtctattg ctactgctgc taactggatg 1380
tggggcttct tgatttcctt ctgcactcca ttcattgtta atgccatcaa cttcaagttc 1440
ggctttgtgt ttactggttg tttactcttt tcgttcttct atgtctactt ctttgtcagc 1500
gaaaccaaag gtttgtcgtt ggaagaagtt gatgagttgt acgctgaagg tattgcacca 1560
tggaagtctg gtgcatgggt tcctccttct gcccaacaac aaatgcaaaa ctccacttat 1620
ggtgccgaag caaaagagca agagcaagtt tag 1653
<210> 35
<211> 2025
<212> DNA
<213> Neurospora crassa
<400> 35
atggcgtcga acccaacgaa caccgcggcc cctacgggtg gccttaccga gaagaagcat 60
gaccgccgtt caacatcgtc cgaatccgtc tcgggaaccg ggtttgcgga acatgcagac 120
cgcaccggca cttttaacca gaacgctcga ctagaggctt caaaaaagat agcgaatcct 180
ttggccggtc taagccctca gcgtctcgag gccatgggag aagaatatgc aatgatggcc 240
ggtctcacca gcgaggagga catcagggcc tttcgactcg gagccagaat cgccggcgat 300
gagagcaact acgacctcat cccggagctt actgaacggg agaaagaggt gttggtgcgc 360
gaaacaactc acaagtggtc taacccaccc atgctttact gggttgttgt catttgctct 420
ctatgcgccg ccgtccaagg aatggacgag acggtcgtca acggcgccca gctcttctac 480
aaggacaagt ttggcattgg tactgatagc cagagagaca cttggcttct gggtctcgtc 540
aactcagcgc cctacctttg ctgtgccttt atcggctgct ggctcactga accgatgaac 600
agaatctttg gcagacgagg caccatcttt gtttcttgca tcatctcagc cgtagcttgc 660
ttccaccagg cctttaccaa cacgtggtgg cacatgttca tcgcccgttt ctacctcggc 720
cttggcatcg gtcccaagtc agccaccacc cccatcttcg ccgccgaatg ctcccctccc 780
aagctccgcg gtgcgctggt catgcaatgg cagatgtgga ccgccttcgg tatcatggtc 840
ggctacattg ccgatctcgc tttctacttc gttcccgatc acggcatcgg cttgggtctg 900
aactggcgtc tgatgatggg ctccgccatg attcccgccg tcatcgtcgt ctgcctcgcc 960
ttcctctgcc cggagtcgcc ccgttggtac ctcagcaagg gccgacacca agacgccttc 1020
ggggcgctct gccgcctgcg tttcgaaaag gtccaagccg cccgcgacct cttctacacc 1080
cacaccctcc tagaagccga gaagcaagcc atgtcgggcg tcaagaaggg taaccgcttt 1140
aaggagctct tcaccgtgcg tcgtaaccgc aacgcggtca ttgcctcgtc gggactcatg 1200
ttcatgcagc agttctgcgg cgtcaacatc atcgcctact actcctcggc ggtcttccga 1260
gacgccggct tcagcgacgt ctcagcactg gccgcctcgc tcgggtttgg cgtcgtcaac 1320
tggctgtttg ccatcccggc catgtacacc atcgacactt tcggccggcg caacctgctg 1380
ctgaccacct tcccgctcat gtccctcttc ctcttcttca ccggcttcag cttttggatc 1440
cccgaggact ccaaagccca catcggctgc atcgcgctag gcatttactt gttcggcatg 1500
gtctactccc ccggtgaagg gccggtgccg tttacttact cggccgaggc ctacccgctg 1560
tacatccggc caatcggcat gtccctcgcc acggcgacta cctggttctt caatttcatt 1620
ctttccatca cctggcctag gatggtcacg gccttcaagc cgcagggcgc gtttggctgg 1680
tatgcagggt ggaatatcat tgggtttctc tttaccctgt tcttggtccc cgagaccaag 1740
ggcaagacgc tggaggagct cgatcacgtg tttgacgtgc cgttgaagaa gttggtcaga 1800
tacggggcgg atcagagctt gtggtttttc cacaggggaa agaatggaaa tggaatgagg 1860
ccgacggcgc ctagtgcgga gatgtatcat ggggatgcgg agcggatgaa cgaggtggtt 1920
agcgggcagc agcttgggga gggtgagagg gagaagaggt ggaacaagga acaagagagg 1980
gaagggggga ttatgggacg aggggatgct gctgggaagg tgtag 2025
<210> 36
<211> 1458
<212> DNA
<213> Neurospora crassa
<400> 36
atgtccgcca tcgtcgtgac cgaccaatac ctcacctact tcaacaaccc ccatgatatc 60
atccaaggag ccatcggctc tgcccttgct gctggctccg tcgtcggttc cgccatcgcc 120
ggtcctcttt ccgacaagat cggtcgtcgt gactccatct ttttcgcctg cttcttctgg 180
ctcattggta cctccgtcca ggttgcctgc aagaactatg gccagctcat cgccggccgt 240
gtgctcaacg gctttaccgt cggcatcact tcctcccagg ttcccgtgta ccttgccgag 300
atcgccaagg cagagaagcg tggttccttg gtcatcatcc agcaactcgc catcgagttt 360
ggtatcttga tcatgtactt tatcggctac ggctgtgcgt cgatcgaggg ccctgcttcg 420
ttccggaccg cttggggcat tcagtttatc ccttgctttt tcctcatggt cggtcttccc 480
ttcttgccta ggtcgcccag atggctggcc aaggtcggta gggaccagga ggccattgct 540
gtcctggcta acatccaggc tgatggcaac gttgatgacc cgagagtcgt tgctgagtgg 600
gaggagattg tcaccgttat gaacgccgag cgtgaggccg gtaagggatg gaggaagttt 660
gtcaagaacg gcatgtggaa gcgaaccatg gctggcatga ctgtacaggc ttggcagcaa 720
ctcgccggcg ccaacgtaat cgtctactac ctaacctaca tcgcccaaat ggccggactc 780
acaggcaacg tcgccatggt gacctcgggc atccaatacg ccgttttcat catcttcacc 840
ggcgtcatgt ggctcttcat cgacaagacc ggtcgtcgca cccttttagt ttacggcgcc 900
ttgggaatgg ccttctgcca ctttgtcgtc ggcggcgtca tgggcgcgca ccacgacaac 960
gttccggacg gcgtcggcgg caacgccaac attgtcatta gcgtgcacaa gggcgcgccc 1020
gccaacacgg tcatcctgtt ctcgtacctg ctcattgtcg tctacgcctt gacgctcgct 1080
cccgtctgct ggatctacgc cgccgaggtc tggtcgttgg gcactcgcgc tacgggcatg 1140
tccatggctg ccatgtccaa ctgggtgttc aactttgcgc tgggcatgtt cacgccgccg 1200
gcgtttgtca atattacgtg gaagctgttt atcattttcg gggtgctttg cgtcacggcg 1260
gcggtctggt tctggttgtt ttacccggag acgtgtggta agacgctgga ggagattgag 1320
atcctgtttg gtgatcaggg tcctaagccg tggaagacaa agaagggcga gtcgagactt 1380
acggcggaga ttgaggctgt caaggcgagg aagacggtgg agcacgagat tgaggtgcat 1440
gagcatgaga aggtttag 1458
<210> 37
<211> 1407
<212> DNA
<213> Pichia stipitis
<400> 37
atgaagtatt ttcaaatctg gaaatcaggc aaacaagtaa gctacgctgt tacattcact 60
tgtgaattgg catttattct ttttggtatt gaacagggta ttattggtaa tcttattaac 120
aaccaggact tcctaaacac ttttggaaac cccaccggta gttatttagg tattatcgtt 180
tctatctata ccttagggtg tttttttggt tgtgttatga acttcttcat tggtgatcga 240
atgggcagaa gaagcaaaat tgcttcctca atgacagtta tcacaattgg tgttgctctt 300
caatgtagtt ccttttcagt tgaacaattg atgattggaa gatttatcac tgggcttgga 360
actggttggg aaacttctac ttgtccaatg tatcaggcag aactttcacc tccaaaagtt 420
agaggacgtt tggtgtgctc agaagcattg tttgttggag ttggtttaat ctatgcatat 480
tggtttgatt atgctctttc tttcacttct ggtcctattg catggagact tcctcttgcc 540
tctcagattg tgttcgcctt tgttgttttc tgtttcactt tcacaatacc cgaatcccct 600
agatacatgt tttacaaagg agagaaagaa gaagccaaaa gaattttatc ttatgtcttt 660
ggaaagccag gagatcatcc tgacattctt aaggaatgga atgatattaa tgatgctgtt 720
attttggaaa cttcagaagg agctttctcg tgggcaaaac ttttcaagcc cgataaggca 780
agaactggat acagagtctt cttggcatac atgagcatgt ttgcgcaaca gttgagtggt 840
gttaatgtag ttaattacta tattacattt gttttgatta acagtgttgg catcgaagac 900
aacttggccc taattcttgg tggtgttgcc gtcatctgtt tcactgttgg ttcattagtt 960
cctactttct ttgctgatag gatgggaaga agattgcctt cagcagttgg agcttttggc 1020
tgtggtgttt gtatgatgct aatttcaatc ttattaagtt ttcaagacaa tccaaagttg 1080
aagaagagca gtggagctgg tgctgtggct ttctttttcg ttttccaact tgtcttcggc 1140
tccactggta attgtattcc atggctgatg atttcagagc ttatccccct tcatgcacgt 1200
gctaaaggat cttcattagc tacatcaagt aactggcttt ggaatttctt tgttgttgag 1260
atcactccaa ctatcattga aaagttgaag tggaaagcat atttgatctt tatgtgctgc 1320
aacttctcct tcgtaccaat gttttacttt ttctttcccg agacaaagaa ccttacttta 1380
gaagccattg acgatttgtt ctcataa 1407
<210> 38
<211> 1653
<212> DNA
<213> Pichia stipitis
<400> 38
atgtcctcac aagatttacc ctcgggtgct caaaccccaa tcgatggttc ttccatcctc 60
gaagataaag ttgagcaaag ttcgtcctca aatagccaaa gtgatttagc ttccattcca 120
gcaacaggta tcaaagccta tctcttggtt tgtttcttct gcatgttggt tgcctttggt 180
ggattcgtat tcggtttcga taccggtaca atttccggtt tccttaatat gtctgatttc 240
ctttccagat ttggtcaaga tggttctgaa ggaaaatatt tgtctgatat cagagtcggt 300
ttgattgttt ccatttttaa cattggttgt gcaattggtg gtattttcct ttctaagata 360
ggagatgttt acggtagaag aattggtatc atttcagcta tggttgtcta cgtcgtcggt 420
attatcatcc agatctcgtc ccaagacaag tggtaccaac ttacaattgg acgtggagtt 480
acaggattag ctgttggtac tgtttcagtg ttgtctccaa tgttcattag tgaaagtgct 540
ccaaagcatt tgagaggtac tttggtatac tgttaccaat tatgtatcac cttaggtatt 600
ttcattggtt actgtgtcac ttatggaacc aaagatttaa atgattcaag acaatggaga 660
gttcctttgg gtttatgttt cctttgggct attttcttag ttgtcggtat gttggctatg 720
cctgaatccc caagattctt aattgaaaag aagagaatcg aagaagccaa gaagtccctt 780
gcaagatcca acaagttatc tccagaagat ccaggtgtct acactgaagt tcaattgatt 840
caggctggta ttgacagaga agctgctgca ggttctgctt catggatgga attgatcact 900
ggtaagccag ctattttcag aagagttatc atgggaatta tcttacagtc tttgcaacaa 960
ttaactggtg tcaactattt cttctattac ggaactacaa tcttccaagc tgttggtttg 1020
caagattcct tccagacttc catcatctta ggtacagtca actttctttc tacatttgtt 1080
ggtatttggg ccattgaaag atttggaaga agacaatgtt tgttagtcgg ttctgctggt 1140
atgttcgttt gtttcatcat ttactccatt attggtacaa ctcatttgtt cattgatgga 1200
gtagtagata acgacaacac ccgtcaactg tctggtaatg ctatgatctt tatcacttgt 1260
ttgttcatct tcttctttgc ctgtacatgg gctggaggtg tttttaccat catttccgaa 1320
tcatatccat tgagaatcag atccaaggca atgtctattg ctactgctgc taactggatg 1380
tggggcttct tgatttcctt ctgcactcca ttcattgtta atgccatcaa cttcaagttc 1440
ggctttgtgt ttactggttg tttactcttt tcgttcttct atgtctactt ctttgtcagc 1500
gaaaccaaag gtttgtcgtt ggaagaagtt gatgagttgt acgctgaagg tattgcacca 1560
tggaagtctg gtgcatgggt tcctccttct gcccaacaac aaatgcaaaa ctccacttat 1620
ggtgccgaag caaaagagca agagcaagtt tag 1653
<210> 39
<211> 1641
<212> DNA
<213> Pichia stipitis
<400> 39
atggaattct ccagtgttga aaaaagtgct gaaactgctt cctatacgtc gcaggtcagc 60
gcaagcggct ctgcaaagac caacagctac cttggcctca gaggccacaa acttaatttt 120
gctgtctctt gttttgctgg tgttggtttc ttacttttcg gttacgatca aggtgtcatg 180
ggttcattgt tgaccttgcc atccttcgaa aacactttcc cggccatgaa ggctagcaac 240
aacgctacct tacaaggcgc cgttattgca ctttatgaaa tcggttgtat gtcttcttct 300
ttagcaacca tttaccttgg tgacagattg ggtagattga agatcatgtt tattggctgt 360
gtaattgtct gtattggtgc tgctttgcaa gcttctgctt tcactattgc tcacttgact 420
gttgctagaa ttatcactgg tttaggtaca ggtttcatca cttctactgt tccagtttac 480
caatcggagt gctctccagc caagaaaaga ggacagttga tcatgatgga aggttctctt 540
atcgcccttg gcattgccat ctcatactgg attgactttg gattttactt tttgagaaac 600
gatggtttgc actcctcggc ttcttggaga gcacctatcg cgcttcaatg tgtcttcgct 660
gtcttgttga tttccacagt cttcttcttc ccagaatctc caagatggtt gctcaacaaa 720
ggtaggaccg aagaagctag agaagttttt tctgctcttt acgacttgcc agccgactct 780
gaaaagattt ctattcaaat tgaagaaatt caagctgcta tagatttaga aagacaagcc 840
ggagaaggtt tcgtacttaa ggaattgttc actcagggcc cagccagaaa cttgcagcgt 900
gtggccttgt catgttggtc tcaaataatg caacaaatca ctggtattaa cattattacg 960
tactatgctg gtactatttt tgaatcatac attggtatga gtccatttat gtcaagaatc 1020
ttggctgcct tgaacggtac tgaatatttc cttgtctctc ttattgcttt ctacaccgtc 1080
gaaagattag gtagaagatt ccttttgttc tggggtgcca tcgccatggc tcttgtcatg 1140
gctggtttaa ctgttaccgt taaacttgcc ggtgaaggca acacccatgc tggtgtcggt 1200
gctgctgttc ttttgtttgc attcaactca ttcttcggcg tctcctggtt aggtggatcc 1260
tggttgttac cacctgaatt gttgtctttg aaattgagag ctcctggtgc tgctttgtcg 1320
accgcttcta actgggcttt taacttcatg gttgtcatga tcactcctgt cggtttccaa 1380
agtattggtt cctacaccta ccttatcttt gctgccatca atttgttgat ggctccggtc 1440
atctacttct tgtatcccga aaccaagggt agatcgttgg aagaaatgga tatcattttc 1500
aaccaatgtc ctgtttggga gccatggaag gttgtccaaa ttgccagaga cctccctatt 1560
atgcactcag aagttcttga ccacgaaaag aatgtcatta ttaaaaaatc tagaatagag 1620
catgtcgaaa acatcagcta a 1641
<210> 40
<211> 1701
<212> DNA
<213> Pichia stipitis
<400> 40
atgcacggtg gtggtgacgg taacgatatc acagaaatta ttgcagccag acgtctccag 60
atcgctggta agtctggtgt ggctggttta gtcgcaaact caagatcttt cttcatcgca 120
gtctttgcat ctcttggtgg attggtctac ggttacaatc aaggtatgtt cggtcaaatt 180
tccggtatgt actcattctc caaagctatt ggtgttgaaa agattcaaga caatcctact 240
ttgcaaggtt tgttgacttc tattcttgaa cttggtgcct gggttggtgt cttgatgaac 300
ggttacattg ctgatagatt gggtcgtaag aagtcagttg ttgtcggtgt tttcttcttc 360
ttcatcggtg tcattgtaca agctgttgct cgtggtggta actacgacta catcttaggt 420
ggtagatttg tcgtcggtat tggtgtgggt attctttcta tggttgtgcc attgtacaat 480
gctgaaattt ctccaccaga aattcgtggt tctttggttg ctttgcaaca attggctatt 540
actttcggta ttatgatttc ttactggatt acctacggta ccaactacat tggtggtact 600
ggctctggtc aaagtaaagc ttcttggttg gttcctattt gtatccaatt ggttccagct 660
ttgctcttgg gtgttggtat cttcttcatg cctgagtctc caagatggtt gatgaacgaa 720
gacagagaag acgaatgttt gtccgttctt tccaacttgc gttccttgag taaggaagat 780
actcttgttc aaatggaatt ccttgaaatg aaggcacaaa agttgttcga aagagaactt 840
tctgcaaagt acttccctca cctccaagac ggttctgcca agagcaactt cttgattggt 900
ttcaaccaat acaagtccat gattactcac tacccaacct tcaagcgtgt tgcagttgcc 960
tgtttaatta tgaccttcca acaatggact ggtgttaact tcatcttgta ctatgctcca 1020
ttcatcttca gttctttagg tttgtctgga aacaccattt ctcttttagc ttctggtgtt 1080
gtcggtatcg tcatgttcct tgctaccatt ccagctgttc tttgggtcga cagacttggt 1140
agaaagccag ttttgatttc cggtgccatt atcatgggta tttgtcactt tgttgtggct 1200
gcaatcttag gtcagttcgg tggtaacttt gtcaaccact ccggtgctgg ttgggttgct 1260
gttgtcttcg tttggatttt cgctatcggt ttcggttact cttggggtcc atgtgcttgg 1320
gtccttgttg ccgaagtctt cccattgggt ttgcgtgcta agggtgtttc tatcggtgcc 1380
tcttctaact ggttgaacaa cttcgctgtc gccatgtcta ccccagattt tgttgctaag 1440
gctaagttcg gtgcttacat tttcttaggt ttgatgtgta ttttcggtgc cgcatacgtt 1500
caattcttct gtccagaaac taagggtcgt accttggaag aaattgatga acttttcggt 1560
gacacctctg gtacttccaa gatggaaaag gaaatccatg agcaaaagct taaggaagtt 1620
ggtttgcttc aattgctcgg tgaagaaaat gcttctgaat ccgaaaacag caaggctgat 1680
gtctaccacg ttgaaaaata a 1701
<210> 41
<211> 1656
<212> DNA
<213> Pichia stipitis
<400> 41
atgagagaag ttggtattct tgatgttgcc catggcaacg ttgtaactat aatgatgaaa 60
gatccagtag tatttttggt gattttattt gcatcccttg gaggtttgct ttttggttat 120
gatcaagggg ttattagtgg cattgtcaca atggaatctt ttggtgcaaa attccccaga 180
atttttatgg atgccgatta caagggttgg tttgtgtcta cttttttgct atgcgcatgg 240
tttggctcta ttattaatac tccaattgtt gataggtttg gaagacgtga ttctatcaca 300
atctcttgtg ttatttttgt cattggttct gcgttccaat gtgctggcat taatacaagt 360
atgttatttg gtgggcgtgc tgttgctggt cttgcagtcg gtcaattaac catggtagtt 420
ccaatgtaca tgtcggaatt ggctcctcca tcggtgagag gtgggttggt tgtaattcag 480
caactttcga ttacaattgg tatcatgatt tcctattggt tggattatgg cactcatttt 540
attggaggta ctagatgtgc tcctagtcac ccataccaag gtgaaacttt taaccctaat 600
gtggatgttc ctccaggtgg ctgctatggt caaagtgatg ccagttggag aattcctttt 660
ggtgttcaga ttgctccagc agtgttgttg ggtattggaa tgatattttt cccaagatct 720
cccagatggt tactctctaa aggtcgcgac gaagaagctt ggagctcttt gaaatatctc 780
agaagaaaga gtcatgagga tcaagtcgaa agagagtttg ctgaaattaa ggcagaggtc 840
gtttatgaag acaagtacaa ggaaaagaga ttccctggta agactggagt tgctttaaca 900
cttactggat actgggatat tcttactact aaatctcact tcaagagagt ttttattgga 960
tcagctgtca tgttcttcca acaattcatt ggctgcaatg caataattta ttacgcacct 1020
acaattttca cacaattggg aatgaactct acaactactt ccttgcttgg tactggtctt 1080
tatggtattg ttaattgtct ttccaccctt ccagcagtgt tcttgatcga tagatgtgga 1140
agaaagactt tgttaatggc aggtgctatt ggaactttta tttccttggt tattgtcggc 1200
gcaatcgttg gcaagtatgg cgatcgttta tctgaattca agacagcagg gagaactgca 1260
attgctttca ttttcattta tgatgtgaat ttctcgtaca gttgggctcc aattggatgg 1320
gttttaccct cagagatttt cccaatcggc atcagatcca atgccatctc cataactacc 1380
tcatctactt ggatgaataa ttttattatt ggcttggtca ctccacatat gttagaaaca 1440
atgaagtggg gcacttacat tttttttgca gcgtttgcta ttattgcgtt ctttttcact 1500
tggcttatca tcccggaaac caagggagtt ccattggaag aaatggatgc cgtgtttggc 1560
gatactgcag cattgcagga aaagaatttg gttaccatta cgtcagtttc tgaatctgac 1620
gccaaggatc gcaactcgat tgaaatgtca gaataa 1656
<210> 42
<211> 1590
<212> DNA
<213> Pichia stipitis
<400> 42
atggcatatc ttgattggtt aacagctaga accaacactt tcgggttgag gggcaagaag 60
ttgagagcct tcatcactgt agtggctgtc actggtttct cattattcgg atatgatcaa 120
gggttgatgt ccggaattat tactgctgat caattcaact ctgagtttcc cgccactaga 180
aataacagta ctatccaagg tgccgtcacc tcctgttacg agcttggttg tttctttggt 240
gctgtgtttg ccttgttaag aggtgaaaga attggaagaa gacctcttgt gctttgtggc 300
tcgcttatta tcatcttggg aacagttatt tctgtaaccg ccttccatcc acactggtca 360
ttaggtcagt ttgttattgg tagagttatc actggtattg gtaatggtat gaatactgcc 420
accattccag tttggcaatc ggaaatgtca agagctgaaa acagaggaag attggtcaac 480
ttggaaggtt ccgttgtcgc tgtgggtaca tgtattgcct actggttgga tttcggtttg 540
tcttatgtcg acaattcagt ttcctggaga tttccagttg ctttccaaat agtgtttgct 600
tccgttttat ttgtgggaat gttgcaattg cccgactctc caagatggtt ggttgctaac 660
cacagaagag cagaggctct tcaagtgttg tctgctttga aagacttgcc cgaagacgac 720
gaagaaattc ttaatgaagc tgaagttatt caggaaagtg tagacaagtt tgctggacat 780
gcttccgtca aggaagtgtt tactggtggt aagacccagc actggcaaag aatggttatt 840
ggatccagca cccaattctt tcagcagttc actggttgta acgctgccat ttactattcc 900
actgtcttgt ttcaagacac tattggttta gaaagaagaa tggcattgat tatcggtggt 960
gttttcgcaa ctgtctacgc cattttcaca attccttcct tcttcttggt cgatactctt 1020
ggacgtagaa acttgttctt gattggtgct atgggacaag gtattgcatt cactatcacc 1080
tttgcctgtt tgattgacga tactgaaaac aacgccaagg gtgccgcagt tggtttattc 1140
ttgtttattt gtttcttcgc cttcaccatc ttgccattgc catgggtata cccaccagaa 1200
atcaatcctt tgagaactag aactatagct tctgcaattt ccacttgtac caactggatc 1260
tgtaactttg ctgttgttat gttcacccct gtctttgtca ctaacaccag atggggagcc 1320
tatcttttct ttgctgtgat gaacttcctt ttcgttccta ttattttctt cttctaccca 1380
gaaacagctg gaagatcgtt ggaagaaatc gatatcatct ttgcgaaggc attcgttgac 1440
aaaagacagc catggagagt tgctgcaacc atgccaaagt tgtccaacca cgaaattgaa 1500
gacgaagcca acagattggg cttgtttgac gatggtacat tcgacaagga agcatttgaa 1560
accaaagaaa acgcatccag cagctcttaa 1590
<210> 43
<211> 1689
<212> DNA
<213> Neurospora crassa
<400> 43
atggcgcctc caaagttcct gggcctctca ggccgaccgc tctctctagc tgtctcgact 60
gtagccacca cgggcttcct tctcttcggc tatgaccaag gtgtcatgag cggcatcatt 120
accgcccccg ctttcaacaa cttcttcaca ccaaccaaag acaactcgac catgcagggt 180
ctcatcactg ccatctacga aattggatgc ttgattggtg ccatgttcgt cctctggacc 240
ggcgatttgt tgggtagacg caggaacatc atggtgggcg ccttcattat ggctctcggt 300
gtcattattc aggttacctg tcaggctgga tccaaccctt ttgctcagct gttcgtcggc 360
agagtcgtca tgggtattgg caacggcatg aacacttcga ccattcccac ttatcaagcc 420
gaatgctcaa agacatcgaa ccgcggtctt ttgatctgca ttgaaggcgg tgtcattgcc 480
tttggtactt tgattgctta ttggatcgac tatggtgcat cttacggtcc cgatgacctc 540
gtttggcgct tccccatcgc tttccagctt ctcttcgcca tcttcatctg cgtccccatg 600
ttttaccttc ccgagtcgcc cagatggctc ctcagccatg gccggaccca agaagctgac 660
aaggtcattg ctgccctccg tggctacgag atcgatggtc ccgagaccat ccaagagcgc 720
aacctcattg ttgactccct gcgtgcctct ggaggtttcg gccaaaagag cactcccttc 780
aaggccctct tcactggcgg caagacccag catttccgtc gtctcttgct cggttccagc 840
tcccagttca tgcagcaagt tggtggttgc aacgccgtca tctactactt ccccattctg 900
ttccaggatt ctattggcga gtcccacaac atgtccatgt tgctgggcgg tatcaacatg 960
atcgtctact ccatcttcgc taccgtttcc tggttcgcca ttgagcgtgt cggtcgtcgt 1020
cgtctgttct tgatcggcac cgttggccag atgctctcca tggtcatcgt cttcgcctgc 1080
ttgatccccg acgaccctat gaaggcccgc ggtgccgcgg tcggtctctt cacttacatt 1140
gcctttttcg gtgccacttg gcttcccctc ccctggctct accccgccga ggttaacccc 1200
atccgcacac gtggaaaggc taacgccgtc tccacctgct ccaactggat gttcaacttc 1260
ctcatcgtca tggtcacccc catcatggtc gacaagattg gctggggaac ttacctcttc 1320
ttcgcggtca tgaacggctg cttccttccc atcatttact tcttctaccc cgagactgcg 1380
aaccgctcgc tcgaggagat cgacatcatc ttcgccaagg gcttcgtcga gaacatgtcg 1440
tacgtcactg ccgccaagga gctgcctcac ctcactgccg aggagatcga gtcctatgcc 1500
aacaagtatg gcctcgtcga ccgcgattcc aacggcgagg gcggcaaccg ccatgacgag 1560
gagaagacgc gcgaccgccc cgaccagagt gacagcgact cccccgctca cgtcgagatt 1620
gatgttgtcg acgagcacgg tgtcgagtcc ggcttcggtg atggtattaa caccaaggaa 1680
acacgttaa 1689
<210> 44
<211> 1626
<212> DNA
<213> Neurospora crassa
<400> 44
atggaattcg agcacgatca ctccgcctcc gacattgaga aggaggccgt cactgtggcc 60
cggccacagg gcgatgtcac ccgcgttgag gctcccgtta ccctcaaggc gtacatgatg 120
tgcgtctttg ccgctttcgg cggtatcttc tttggctacg attcaggtta catctctggt 180
gtcatgggca tgaagtactt tatcgaaacc atcaacggac ccggcgccac cttcctgcca 240
tccaaggaaa agtcgctcat cacctccatt ctctctgccg gaaccttctt tggcgccctc 300
atgggcggtg atctcgctga ctgggttggc cgtcgtccta ccatcatctt cggctgcctc 360
gtcttcatcg tcggtgttgt tctccagact gcctcccaga gcttgggtct cattgtggcc 420
ggccgtctcg tcgctggttt cggtgtcggt ttcgtctcgg ccattatcat cctgtacatg 480
tctgagatcg cgccccgcaa ggtccgcggt gctatggtgt cgggctacca gttctgcatc 540
tgcctgggtc tgctcctggc ctcgtgcgtt gactacggca cccagaaccg caccgacagc 600
ggctcttaca gaatcccgat tggtctccag atggcctggg ccctcattct tgctactggt 660
atctttttcc ttcctgaatc ccctcgcttc ttcgtcaaga agggcaagct cgacaaggcc 720
gccggcgtgc tctcccgcct gcgcgaccag ccgctcgatt ccgactacgt cagggacgaa 780
cttgccgaga tcgttgccaa ccacgaattc gaaatgaccg tcgtccccta cggcaactac 840
ttccagcagt gggccaactg cttccgcggc tccatctggc agggtggttc ttacctccgc 900
cgcaccattc tcggcacttc gatgcagatg atgcagcagt ggacgggaat caactttatc 960
ttttactttg gaaccacctt cttccagcag ctcggcacca ttgacaaccc cttcctgatg 1020
tctctggtca ctactcttgt caacgtctgc tccaccccca tctccttcta caccatggag 1080
aagctcggcc gtcgtaccct cctcatctgg ggcgctctcg gcatgctgat ctgcgagttt 1140
atcgtcgcca tcgttggtac ctgcaggccg gatgatacca tggccatcaa ggccatgctc 1200
gccttcatct gcatctacat cttcttcttt gctaccacct ggggccctgc ttcctgggtc 1260
gtcatcggcg aggttttccc tcttcccatt cgtgccaagg gtgttgccct ttccaccgcc 1320
tccaactggc tctgtaactg catcatcgcc gtcatcactc cctacatggt cgacgaggac 1380
aagggcaacc tgggccccaa ggtgttctac atctggggtg gcctctgcac ctgctgcttc 1440
atctacgcct acctgcttgt gcccgagacc aagggcctca cgctcgagca ggtcgaccaa 1500
atgctttccg agtctacccc ccgcacctcg accaagtgga agcctcacac cacttatgct 1560
gctgagatgg gcatgaccga gaagactgtt gctggccacg ctgagaaccg cagcgatagc 1620
gagtaa 1626
<210> 45
<211> 1614
<212> DNA
<213> Neurospora crassa
<400> 45
atgggtcttt cgataggaaa taggatcctc cggaaaattg tcaaaaatga ggccatggca 60
gaagatcccc cagagatcta tggctggcgt gtctatctcc tagcgtgctc tgcctgcttc 120
ggcgccatgt ctttcggctg ggattcctcc gtcatcggcg gcgtcatcga actcgaaccc 180
tttaaacacg actttggctt catcggcaac gataaagcca aggccaacct gggcgccaat 240
atcgtctcta ccctccaagc cggctgcttc ctcggtgcgc tgatcgcctc acctataacc 300
gatcgcttcg gccgcaagtg gtgtctcatc gctgtctccc tggtcgtcat catcggtatc 360
atcatgcaag ccgccgcctc aggcaacctc gcacccatgt acattggccg tttcgtcgcc 420
ggcgtgggcg tcggcgccgc cagctgcatc aaccccgtct ttgtgtctga gaacgctccc 480
cgctcgatcc gcggtctgtt gacgggcctc taccaactct tcattgtcac cggcggcatg 540
atcgcatttt ggatcaacta ctccgtctct ctgcacttca agggcaaatc catgtacatc 600
ttcccgctcg ccatccaagg tcttcccgcc ggccttttgt gcgtctgcat gctcctctgc 660
cacgaaagcc cgcgctggct ggcccgtcgt gaccgatggg aagaatgcaa gtctgtgctg 720
gcgcgcatcc gcaacctccc cccagaccac ccgtacatcg tcgacgagtt ccgcgagatc 780
caggaccagc tcgaacagga gcgtcgtctc cagggcgacg ccacttactg ggacttgacc 840
cgcgatatgt ggaccgtcgc cggcaaccgc aagcgcgccc tgattagtat tttcttgatg 900
atctgccagc aaatgacggg caccaacgcc atcaacacgt acgcgcctac catcttcaag 960
aacttgggta tcaccggcac gtcgactagc ttgtttagta ccggcatcta tggtattgtc 1020
aaggtcgtta gctgcgtcat tttcttgctg ttcttggccg actcgctggg tcgtagacgt 1080
tcgctgctgt ggacgtcgat tgcgcagggt cttgctatgt tttatattgg cctttatgtc 1140
cgcatctcgc cgccgattga tggccagccg gtgccgcctg cgggttatgt agcgttggtg 1200
tgcatctttc tgtttgccgc tttcttccaa tttggctggg gtcctgcctg ctggatctac 1260
gcctcggaaa tccccgccgc ccgcctgcgc tccctcaacg tgtcctacgc cgccgcgacg 1320
cagtggctgt tcaatttcgt cgtggcccgc gccgtgccta ctatgctggt cacggtcggc 1380
ccccacggtt acggcaccta cctcatcttt ggcagcttct gcctcagcat gtttgtcttt 1440
gtctggttct tcgtgcccga gacaaagggt atctcgcttg agcacatgga tgagctgttt 1500
ggcgttactg atgggcctgc cgctgagaag tcgtcggtgc atggtggaga tgatgtcggg 1560
tcggagatgg ggaaggggga tcagaagtcg aagcatgtgg aggtttatgt ttaa 1614
<210> 46
<211> 1587
<212> DNA
<213> Pichia stipitis
<400> 46
atgtcttcgt tattgactaa cgaatacttc aaagactact accacaaccc gactcctgtt 60
gaagtgggta ctatgattgc tatcttagag atcggcgcac ttttttcctc cttcatagct 120
ggaagagtag gtgacatcgt tggcagaaga agaaccatta gatacgggtc tttcattttt 180
gtagtaggcg gtcttgtaca agctacttcg gtcaatattg tcaatctctc actaggaaga 240
ttgattgccg gtattgccat tggctttttg acaaccatca tcccatgcta ccagtctgaa 300
atcagccccc cagacgatag aggtttctat gcctgtttgg agttcaccgg aaatatcatt 360
ggatatgcta gtagtatttg ggtagactac gggttttcat ttttagacaa tgatttcagc 420
tggaggagcc cattgtatgt tcaggttgtt attggctcca tgttatttat tggttcattc 480
cttattgtag aaacccctag atggctcttg gatcacaacc atgatatcga aggcatgatt 540
gtcatttctg acttgtatgc agatggtgat gtggaagacg atgatgctat tgctgagtac 600
agaaacataa aggaaagtgt cttgatagcc agagttgaag gcggagagag atcgtaccag 660
tatttgttca ccaaatatac caagagactt tctgtggcat gcttttcgca aatgtttgcc 720
cagatgaatg gtataaacat ggtatcttac tatgctccta tgatcttcga atctgctggc 780
tgggttggta gacaagctat cttgatgact ggtatcaact ccattatcta catctttagt 840
accattcctc catggtactt agttgattct tggggcagaa aacctttgct tttatctgga 900
tctgtgctca tgggtgttcc gctcttaacc attgcttgtt cgttattctt aaacaacaca 960
tacacacccg gggttgtggt tggcagtgta atcgtattca atgctgcttt tggatacagt 1020
tggggtccaa ttccttggct catgagcgaa gtgttcccta actcagttag atcaaaaggt 1080
gctgccatgt ctactgcaac caactggctc tttaacttta ttgttggaga gatgacacct 1140
attttgttgg atacaattac ctggagaact tacttgatcc cggcaacttc gtgtgtatta 1200
tcgttttttg ctgttggatt tttatttcca gagaccaagg gtttagcatt ggaggatatg 1260
ggctccgtat tcgatgataa ttcgtcaata ttttcatatc actcaacttc ttccactggg 1320
tatggtgcga ccgagtctaa cagtaatgcc aggagagcaa gtgtcatctc ttcagaaaac 1380
taccaggata gtttgcatca gacagcggct tcattggcta gaaatccttc aagcatgagg 1440
cctgattacg atggcataat cacaggagct gctacccttt cgccagtacc accattaaaa 1500
ccaataaaca tttccagcaa tattccgcag gaaattgaac caccaacctt tgatgaaatc 1560
tttaagtaca agttgaatga gatggaa 1587
<210> 47
<211> 1257
<212> DNA
<213> Pichia stipitis
<400> 47
atgacttttg cagttaactt gtatgtgttt gcagttggta gagtgctttc tggggtgggt 60
gtaggagttc tatcgactat ggtgccgtcc tatcaatgcg aaattagtcc cagcgaagaa 120
agaggcaagt tggtgtgtgg agagttcacg ggaaatatca ctggttatgc tctcagtgta 180
tgggccgatt acttctgcta ctttattcaa gatataggtg atgcaaggga gaagcctcat 240
agcttctttg cccacttgtc ctggcgattg cctctattca tccaggtggt gatagcggct 300
gttctctttg ttgggggatt ttttattgtc gagtcacctc gttggttatt agatgtagac 360
caggaccaac aaggattcca tgtattagcg ttgctctatg attcacatct agatgataac 420
aaaccacgtg aagagttctt tatgatcaaa aactccatct tgttagaaag agaaactaca 480
cctaagagcg aacgaacttg gaaacatatg ttcaagaact acatgacccg agtgcttata 540
gcttgttcag cacttggctt tgcacagttc aacggcataa atatcatttc gtactatgcc 600
cccatggtat ttgaagaagc aggcttcaac aactccaagg ctttacttat gacaggcatc 660
aactctatag tatattggtt cagtacgatt cctccgtggt ttctcgtgga tcattggggt 720
agaaagccaa ttttgatatc cgggggttta tctatgggaa tatgtattgg tttgattgcg 780
gtggtaattc tactagacaa gtcgttcaca ccgtctatgg ttgcggtatt ggtgataatc 840
tacaatgcat cttttggcta cagttggggt cctatcggat tcttgatccc gccggaggtg 900
atgccattgg cagttagatc gaaaggtgtt tctatttcta cggctacaaa ctggtttgcc 960
aattttgttg tgggtcagat gacgccaatt ctacagcaga gattgggctg gggaacttat 1020
ctattcccgg ctggtagttg tatcatctcg gtgatagtgg tgattttctt ctatccagag 1080
acaaagggtg cagagctaga ggatatggac tctgtgttcg agagctttta caactacaag 1140
tctccgttca agatttcacg aaagagacac cagaatgatg gccaggcgta ccaaagggta 1200
gagaacgata tccgccacaa cgatgtagaa atggacgatt tggacgattt ggactaa 1257
<210> 48
<211> 1757
<212> DNA
<213> Neurospora crassa
<400> 48
atggaattcg gtggcggagg cggctccggc gcagctggtt tttatgatgc ggctcttcag 60
aggcgtgagg cagtgatggg gaagagtggc cctgcagcac ttgtcaagaa cttccgggtc 120
ttttctattg catgcttcgc atgtatcggt ggtgtgctct atgggtacaa ccaagggatg 180
ttttcgggtg tcctcgccat gccagccttt cagaaacaca tgggcgaata cgatccgata 240
gacgagaacg cgagtcagac aaagaagggc tggctaaccg caattcttga gctcggtgct 300
tggcttggta cgcttctgtc tgggttcatg gcagaggttc tctcgagaaa gtacggtgtg 360
ctagtggcgt gcttggtttt catgctgggt gtggtcatcc aagccacgtc tatctctgga 420
ggacatgaga ccattcttgc cggacggttt atcacgggta tgggtgtcgg atccttagcc 480
atgatcattc ccatttacaa ctcggaagtt gcaccacctg aggtccgtgg agctcttgtt 540
gctctccagc agttggctat ctgcttcggt atcatggtca gcttctggat tgactacgga 600
accaactata tcggcggcac caagctcgag acccaatccg acgccgcctg gcttgtaccc 660
gtctgcctgc aactcgcccc tgctctcatt ctgtttttcg gcatgatgtt catgcccttc 720
tccccacgct ggctcatcca ccatggccgc gaggcggaag ctcgaaagat cctctccacc 780
cttcgcggtc taccccaaga ccacgagctt gtcgagctcg agttcctcga aataaaggct 840
cagtctctct tcgaaaaacg cagcattgcc gagttgtttc ccgaattgcg cgagcagact 900
gcctggaata cctttaagct ccagtttgtc gccatagaga agcttttccg gacaaaggca 960
atgttccgac gcgttgtcgt ggcaaccgta accatgttct tccagcagtg gtccggcatc 1020
aatgcgattc tctactacgc cccgcaaatc ttcaagcagc ttggactgag cggtaacaca 1080
acctcactcc tggctacggg tgtagtaggc atcgtcatgt tcatcgcaac ggttcctgcc 1140
gtgctgtgga tcgaccgtgt tggtcgcaag cccgtgctta ctatcggtgc cctcgggatg 1200
gctacctgcc atatcatcat cgctgtcatt gttgccaaga acgtggacca atgggagact 1260
cataaggctg ctggatgggc tgctgtagcc atggtctggc tattcgtcat tcactttgga 1320
tattcatggg gtccatgtgc ctggatcatt gttgctgaga tctggccgtt gagtacgagg 1380
ccatatggtg tctctctagg agcttcgagc aactggatga acaactttat cgtcggtcag 1440
gtcacgccgg atatgttaaa ggcgatcccg tacggaacgt atatcatctt cgggttgttg 1500
actatatggg tgccgccttt atttggttct ttgtgccgga aacgaagaga ttaaccttgg 1560
aagagatgga catgatcttc ggatccgaag gcactgcaca agccgacaat gagcgcatgg 1620
aggagatcaa tgctgagatt ggtcttaccc gattcctgca aggtggtagt ggtgcaaacc 1680
aaggtgctgc tgatggaagc gatactggtt atgatgcgga gaagggcaag agcgaacact 1740
attctcagca tgtctaa 1757
<210> 49
<211> 1584
<212> DNA
<213> Neurospora crassa
<400> 49
atgggcttgt cactcaagaa gcctgaaggt gtgccgggca agtcatggcc cgccattgtc 60
attggcttgt ttgtcgcctt tggtggtgta ctctttgggt atgacactgg cactattggc 120
ggtatccttg ctatgcccta ttggcaagat ttgttttcga caggttacag aaacccagag 180
catcacttgg acgttaccgc gtcgcagtct gccactatcg tctccattct gtctgctgga 240
accttctttg gcgctcttgg tgccgctccc cttgccgact gggctggacg acgcttgggc 300
cttattctgt cgtcgtttgt gtttatcttc ggtgtcatcc tgcagaccgc agccgtcagc 360
attcctcttt ttctggctgg ccgattcttt gctggattgg gagttggtct catatcggca 420
accatccccc tctatcaatc cgagactgcc ccgaaatgga ttcgtggtgt catcgtcggg 480
tcctatcagc tagccattac catcggtctt cttcttgcct ccattgtcaa caatgccacg 540
cataacatgc agaacaccgg ctgctatcgc atccccatag ctgtccaatt tgcatgggcg 600
atcatcctga tcgttggcat gatcattctt cccgaaactc cacgctttca tatcaagaga 660
gacaatctcc cagccgccac taggtctcta gctatcctcc gccgtctgga gcagaaccat 720
ccagcgatca tcgaagagct ttccgagatc caagccaatc atgaatttga gaagagcctc 780
gggaaggcga cctacttgga ctgcctcaag ggcaatttac tcaagcggct ccttactggc 840
tgttttctcc agagcctgca gcagttgact ggcatcaact ttatcttcta ctacggcaca 900
cagttcttca aaaactccgg attctcagac tcgtttctga tatccttgat cactaatctt 960
gtcaatgtcg tgtcgaccct tcccggactc tacgccatcg acaaatgggg ccggaggcct 1020
gttttactct ggggagctgt tgggatgtgt gtctgccagt tcatcgttgc tattcttggg 1080
acaacaacga caagtcaaga tgcaagcgga atgatcattg tgcataatct cgccgcacag 1140
aaagcagcta ttgcattcat ctgcttctac atctttttct tcgctgcatc ttggggtcca 1200
gttgcctggg tcgttacagg cgagatcttc ccccttaaag tccgcgccaa gtcgctctcc 1260
ataactacag cgtcgaattg gctgctcaac tgggccattg cttacagcac accttacctt 1320
gtcaactacg gccctggcaa tgcgaacctg cagtccaaga tcttcttcgt ctggggcgga 1380
tgctgcttca tctgcatcgc attcgtttac ttcatgatct atgagacaaa aggtctcaca 1440
ctggagcagg ttgacgagct atatgaagag gtctcggatg ccaggaagag tattggttgg 1500
gtgccgacca tcactttccg ggagatccgg gaggaaaaga aagtaaggga tccagttgtt 1560
gatatcactg aagaggcagc ttga 1584
<210> 50
<211> 1968
<212> DNA
<213> Neurospora crassa
<400> 50
atgggccaca atccagacct ggacagtagc ggcactgccg gagaacccaa aggtgtcacc 60
ggctcgcaca ttgaacaaac ctcgtccaac ctcgaagcca acatcaacct cgaagccaag 120
ctcaagaacc cgctcgacgg cctttcccgc gtcgagctcc tgtcacgcgt cgagaccttt 180
tgcgccgaaa agaacctaac cgagcacctc cctcttttcc gtaagggagc actcatcgcc 240
cagtccccgg acagctatgc gtccatctcg ggcccggaag ccttggacga tgaggagaag 300
gcagtacttt tgaaggaggt cgaacacaag tggcggctgc cggcaagact gttcctgacg 360
attgctactt gctcgatcgg tgctgctgtc caaggttggg atcagacggg cacgaatggc 420
gcgaatatct tctttcccaa ctattacggt atcggaggcg acactgcgag ggagaagttg 480
cttgtcggat tgatcaatgc tgggccctat attgggagcg cattcatcgg ttgctggctt 540
tctgatccca tcaacaactg gattggtcgt cgtggtgtta tctttgtctc tgctcacttc 600
tgtatctggc ccgtcatcgg ttctgctttc tgtcacacat ggccccagca actggcctgc 660
cgtctgctga tgggtatcgg tatgggtgtg aaggcatcaa cggtgccgat ctatgccgcg 720
gaaaactcgc ctgcttctat tcgaggtgcg ctggtcatgt catggcagat gtggacagcc 780
ttcggcatct tcttgggcac tgcctttaac cttgccgtct tccacgccag ctccaacgtt 840
aactggcgcc tcatgctcgg tgcccccttc attcccgccg tacccctgct tctgctcatc 900
tatctttgcc ccgagtcccc gcgctggtac atgaagaagg gccgctaccc agaagcctgg 960
aaatccatgg tcaagctgcg caaccacccc atccaagttg cccgcgacat gttctacatc 1020
cactcgcaat tggaagtcga gcaccagctc ctcgccggct ccaactatgc caagcgcttc 1080
gtcgagctct tcaccgtccc tcgtgttcgc cgcgccaccc tcgccgcttt caccgtcatg 1140
attgcccagc agatgtgcgg aatcaacatc atcgcctttt acagcaccac catcttcaag 1200
gattccggct ccaccgaatt ccaagccctg ctttcctcct tcggcttcgg tctagtcaat 1260
tggctctttg ccttccccgc cttctggact atcgacactt ttggccggcg ctctctgctt 1320
ctttttacct tcccgcaaat gatgtggacc ctgctagcag ccggcctctt caccttgctc 1380
gacatgggtc ccgcccggac cgggctcgtc gccttattcg tcttcctctt cgccgcgttc 1440
tactcacccg gtgaaggtcc tgtccccttc acctactcgg ccgaagtctt ccccctctct 1500
cacagagaag taggcatggg cttcgccgtc gccacctgcc tcttctgggc atctgttttg 1560
ggtattacct tccccttctt gcttgactct ctcggcaccg tcggcgcctt tggtctgtac 1620
gcgggcttca acctagtggc gtttattgcc atcttcttgg tcgtgccgga gacgaagcag 1680
aagacgctcg aggagttgga ttatgtcttt gctgtgaaga cgagcaagtt catgtcgtat 1740
cagtgcacca aggcgctgcc gtggttcatc aagaggtggg tgttttggca gaggaatgca 1800
aagctggagc cactgtatga gtttgatcgg atcaaggagg cggagaagga gaggagagca 1860
gaggaggaga gaagggcaaa ggagacggga acgatcacct ctactgctac aggagctgag 1920
ttggatgaga agaagggact gagtcatgtt aatgctccta attcttag 1968
<210> 51
<211> 1725
<212> DNA
<213> Saccharomyces cerevisiae
<400> 51
atggcagttg aggagaacaa tatgcctgtt gtttcacagc aaccccaagc tggtgaagac 60
gtgatctctt cactcagtaa agattcccat ttaagcgcac aatctcaaaa gtattctaat 120
gatgaattga aagccggtga gtcagggtct gaaggctccc aaagtgttcc tatagagata 180
cccaagaagc ccatgtctga atatgttacc gtttccttgc tttgtttgtg tgttgccttc 240
ggcggcttca tgtttggctg ggataccggt actatttctg ggtttgttgt ccaaacagac 300
tttttgagaa ggtttggtat gaaacataag gatggtaccc actatttgtc aaacgtcaga 360
acaggtttaa tcgtcgccat tttcaatatt ggctgtgcct ttggtggtat tatactttcc 420
aaaggtggag atatgtatgg ccgtaaaaag ggtctttcga ttgtcgtctc ggtttatata 480
gttggtatta tcattcaaat tgcctctatc aacaagtggt accaatattt cattggtaga 540
atcatatctg gtttgggtgt cggcggcatc gccgtcttat gtcctatgtt gatctctgaa 600
attgctccaa agcacttgag aggcacacta gtttcttgtt atcagctgat gattactgca 660
ggtatctttt tgggctactg tactaattac ggtacaaaga gctattcgaa ctcagttcaa 720
tggagagttc cattagggct atgtttcgct tggtcattat ttatgattgg cgctttgacg 780
ttagttcctg aatccccacg ttatttatgt gaggtgaata aggtagaaga cgccaagcgt 840
tccattgcta agtctaacaa ggtgtcacca gaggatcctg ccgtccaggc agagttagat 900
ctgatcatgg ccggtataga agctgaaaaa ctggctggca atgcgtcctg gggggaatta 960
ttttccacca agaccaaagt atttcaacgt ttgttgatgg gtgtgtttgt tcaaatgttc 1020
caacaattaa ccggtaacaa ttattttttc tactacggta ccgttatttt caagtcagtt 1080
ggcctggatg attcctttga aacatccatt gtcattggtg tagtcaactt tgcctccact 1140
ttctttagtt tgtggactgt cgaaaacttg ggacatcgta aatgtttact tttgggcgct 1200
gccactatga tggcttgtat ggtcatctac gcctctgttg gtgttactag attatatcct 1260
cacggtaaaa gccagccatc ttctaaaggt gccggtaact gtatgattgt ctttacctgt 1320
ttttatattt tctgttatgc cacaacctgg gcgccagttg cctgggtcat cacagcagaa 1380
tcattcccac tgagagtcaa gtcgaaatgt atggcgttgg cctctgcttc caattgggta 1440
tgggggttct tgattgcatt tttcacccca ttcatcacat ctgccattaa cttctactac 1500
ggttatgtct tcatgggctg tttggttgcc atgttttttt atgtcttttt ctttgttcca 1560
gaaactaaag gcctatcgtt agaagaaatt caagaattat gggaagaagg tgttttacct 1620
tggaaatctg aaggctggat tccttcatcc agaagaggta ataattacga tttagaggat 1680
ttacaacatg acgacaaacc gtggtacaag gccatgctag aataa 1725
<210> 52
<211> 1908
<212> DNA
<213> Pichia stipitis
<400> 52
atgagtgctg acgaaaaagt cgctgctgcc ggccaggacg gcttgtttga acacaacagt 60
tccacttcga gcatcgagga caagaagccc tccaagagct ccgatgtcga ttccgtgaac 120
tcgcaattag tagacaactc ggtagagggc aacatcttgt cccagtacac cgaaagtcag 180
gtgatgcaga tgggtagaag ctatgccacc aagcacggct tggacccaga attgttcgcc 240
aaggcagctg ctgttgccag aactcctctt ggtttcaact ccatgccctt cttgacagag 300
gaagagaagg ttggtttgaa tgccgaagcc actaataagt ggcacattcc acccagattg 360
atcggggtta ttgccttggg ttctatggcc gctgctgtgc agggtatgga cgaatcggtc 420
attaacggtg ccaacttgtt ctaccccaag gctttcggag tcgacaccat gcacaattcg 480
gacttgattg aaggtttgat caatggtgct ccttaccttt gctgtggtat tctttcctgt 540
tggttgtctg acgcttgtaa ccgtcgtctt ggtagaaaat ggaccatttt ctggtgttgt 600
gtcatttctg ccatcacctg tgtctggcaa ggtcttgtca acaactggta ccatttgttc 660
attgctcgtt tcttccttgg atttggtgtt ggtatcaagt ccgccactgt tcctgcctac 720
tctgccgaat gtactcctaa acacatcaga ggttcgttag tcatgttgtg gcaattcttc 780
acagctgttg gtattatgtt tggttatgtt gcttccttgg ctttctacaa tgtcggagat 840
agaggaatcc attacgggtt gaactggaga ttgatgcttg gttcggccgc tattcctgct 900
gtcatcatct tgttccaaat tcctttcgct cctgaatctc cacgttggtt aatgggtaag 960
gacagacacc ttgaagcctt tgagtccttg aagcaattga gatacgaaga acttgctgct 1020
gctcgtgact gtttctacca gtacgtcttg ttagctgaag aaggttctta caagatccca 1080
accctcacca gatttaagga aatgttcacc aagagaagaa acagaaacgg tgccatcggt 1140
gcatttattg tcatgttcat gcaacagttc tgtggtatca acgtcattgc ttactactct 1200
tcgtctatct ttgtccaatc tggtttctct caaacttctg ctttgatcgc ttcttggggt 1260
ttcggtatgc ttaacttcac ctttgccatt cctgccttct tcacaatcga tcgtttcggt 1320
agaagatcct tattgttggt taccttcccc ttgatggcta ttttcttatt gattgccggt 1380
ttcggtttct tgataaacga agaaacaaac tccaagggaa gattgggaat gatcatcatc 1440
ggtatctata tgttcaccat ctgttactct tccggtgaag gtccagttcc tttcacctac 1500
tctgccgaag ccttcccatt gtacatcaga gacttgggta tgtcttttgc tactgccacc 1560
tgttggactt tcaacttcat cttggccttc acctggaaca gattggtcaa tgcattcaca 1620
tctactggtg ccttcggctt ctacgctgct tggaacatca ttggtttctt cttggtctta 1680
tggttcttgc cagaaaccaa gggcttgacc ttggaagaat tggacgaagt cttcgccgtt 1740
tccgccgtcc aacacgccaa gtaccaaacc aagagtttga tcaacttcat ccaaagatac 1800
gttttacgtt ccaaggtggc tccattgcct ccattgtacg accaccagag attggctgtc 1860
accaacccag aatggaacga caagccagaa gtctcttatg tcgagtag 1908
<210> 53
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 53
atatatgagc tcgtgagtaa ggaaagagtg aggaactatc 40
<210> 54
<211> 50
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 54
atatatacta gttgttttat atttgttgta aaaagtagat aattacttcc 50
<210> 55
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 55
atggatccaa aaatgtcgtc tcacggctcc 30
<210> 56
<211> 60
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 56
atgaattcct acaaatcttc ttcagaaatc aatttttgtt cagcaacgat agcttcggac 60
<210> 57
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 57
atactagtaa aaatgggcat cttcaacaag aagc 34
<210> 58
<211> 64
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 58
gcatatcgat ctacaaatct tcttcagaaa tcaatttttg ttcagcaaca gacttgccct 60
catg 64
<210> 59
<211> 42
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 59
tattaaatcg atggtagtgg tagtgtgagc aagggcgagg ag 42
<210> 60
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 60
tattaagtcg acctacttgt acagctcgtc catgcc 36
<210> 61
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 61
gcatggatcc atgtcgtctc acggctcc 28
<210> 62
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 62
tataatgaat tcagcaacga tagcttcgga c 31
<210> 63
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 63
tattaaacta gtatgggcat cttcaacaag aagc 34
<210> 64
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 64
ttataagaat tcagcaacag acttgccctc atg 33
<210> 65
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 65
gcatactagt aaaaatgtct cttcctaagg atttcctct 39
<210> 66
<211> 55
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 66
atactgcagt taatgatgat gatgatgatg gtccttcttg atcaaagagt caaag 55
<210> 67
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 67
Pro Glu Ser Pro Arg Phe
1 5
<210> 68
<211> 4
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 68
Pro Glu Ser Pro
1
<210> 69
<211> 4
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 69
Pro Glu Ser Pro
1
<210> 70
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> GS-linker
<400> 70
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10
<210> 71
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 71
gacatcgatg acatatgcgc taccgcaac 29
<210> 72
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 72
gtgcacgtcg gacccgcaga ttcc 24
<210> 73
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 73
ggaatctgcg ggtccgacgt gcac 24
<210> 74
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 74
cagaagattt aaggatcctg aacgtaga 28
<210> 75
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 75
gacatcagtg acatatgtcg ccttcc 26
<210> 76
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 76
cctggattga ggatcctgaa cgtata 26
<210> 77
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 77
gacatcgatg acatatggct tccgcaac 28
<210> 78
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 78
ccagaagtat tgagaattct gaacgtaga 29
<210> 79
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 79
gacatcgatg acatatggcg actctgc 27
<210> 80
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 80
ggatacagaa tgaggatcct gaacgtaga 29
<210> 81
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 81
taatacgact cactataggg 20
<210> 82
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 82
gctagttatt gctcagcgg 19
<210> 83
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 83
cctatcgtca ttacctcacg tgacgagggg cggctg 36
<210> 84
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 84
cagccgcccc tcgtcacgtg aggtaatgac gatagg 36
<210> 85
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 85
ccttcgaaac ggctacaaac cccaagacg 29
<210> 86
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 86
gcttgtcatc acatcacgtt cagagagccg tctg 34
<210> 87
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 87
cagacggctc tctgaacgtg atgtgatgac aagc 34
<210> 88
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 88
gcatttgaga cgtcaacaga tcccaagagc 30
<210> 89
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 89
cctattgtca tcacttcacg tgacgagggc cgcttg 36
<210> 90
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 90
caagcggccc tcgtcacgtg aagtgatgac aatagg 36
<210> 91
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 91
cctttgagac tgccacaaac cctaagaccg gtg 33
<210> 92
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 92
atggatccaa aaatgtcgtc tcacggctcc 30
<210> 93
<211> 60
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 93
atgaattcct acaaatcttc ttcagaaatc aatttttgtt cagcaacgat agcttcggac 60
<210> 94
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 94
atactagtaa aaatgggcat cttcaacaag aagc 34
<210> 95
<211> 64
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 95
gcatatcgat ctacaaatct tcttcagaaa tcaatttttg ttcagcaaca gacttgccct 60
catg 64
<210> 96
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 96
gcatactagt aaaaatgtct cttcctaagg atttcctct 39
<210> 97
<211> 55
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthesized Construct
<400> 97
atactgcagt taatgatgat gatgatgatg gtccttcttg atcaaagagt caaag 55
Claims (119)
- 하기 단계를 포함하는, 세포 내로 셀로덱스트린(cellodextrin) 수송(transport)을 증가시키는 방법:
- 숙주 세포(host cell)를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인(transmembrane) α-헬릭스 1(α-helix 1), α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드(polypeptide)를 코딩하는(encoding) 재조합 폴리뉴클레오티드(recombinant polynucleotide)를 포함하고, 트랜스멤브레인 α-헬릭스 1 은 SEQ ID NO : 1 을 포함한다); 및
- 재조합 폴리뉴클레오티드가 발현되는 배지(medium)에 세포를 배양하는 단계
(재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로 셀로덱스트린의 수송이 증가되었다).
- 하기 단계를 포함하는, 세포 내로 셀로덱스트린의 수송을 증가시키는 방법:
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 2 은 SEQ ID NO : 2 을 포함한다) ; 및
- 재조합 폴리뉴클레오티드가 발현되는 배지에 세포를 배양하는 단계
(재조합 폴리뉴클레오티드가 포함되지 않는 세포와 비교하여, 재조합 폴리뉴클레오티드의 발현으로 세포 내로 셀로덱스트린의 수송이 증가되었다).
- 하기 단계를 포함하는, 세포 내로 셀로덱스트린 수송을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 2 및 트랜스멤브레인 α-헬릭스 3 을 연결하는 루프(loop)는 SEQ ID NO : 3 을 포함한다) ; 및
- 재조합 폴리뉴클레오티드가 발현되는 배지에 세포를 배양하는 단계
(재조합 폴리뉴클레오티드가 포함되지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로 셀로덱스트린의 수송이 증가되었다).
- 하기 단계를 포함하는, 세포 내로 셀로덱스트린 수송을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 5 는 SEQ ID NO : 4 를 포함한다) ; 및
- 재조합 폴리뉴클레오티드가 발현되는 배지에 세포를 배양하는 단계
(재조합 폴리뉴클레오티드가 포함되지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로 셀로덱스트린의 수송이 증가되었다).
- 하기 단계를 포함하는, 세포 내로 셀로덱스트린 수송을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 6 는 SEQ ID NO : 5 를 포함한다) ; 및
- 재조합 폴리뉴클레오티드가 발현되는 배지에 세포를 배양하는 단계
(재조합 폴리뉴클레오티드가 포함되지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로 셀로덱스트린의 수송이 증가되었다).
- 하기 단계를 포함하는, 세포 내로 셀로덱스트린 수송을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 6 및 트랜스멤브레인 α-헬릭스 7 사이의 서열은 SEQ ID NO : 6 을 포함한다) ; 및
- 재조합 폴리뉴클레오티드가 발현되는 배지에 세포를 배양하는 단계
(재조합 폴리뉴클레오티드가 포함되지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로 셀로덱스트린의 수송이 증가되었다).
- 하기 단계를 포함하는, 세포 내로 셀로덱스트린 수송을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 7 은 SEQ ID NO : 7 을 포함한다) ; 및
- 재조합 폴리뉴클레오티드가 발현되는 배지에 세포를 배양하는 단계
(재조합 폴리뉴클레오티드가 포함되지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로 셀로덱스트린의 수송이 증가되었다).
- 하기 단계를 포함하는, 세포 내로 셀로덱스트린 수송을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 10, 트랜스멤브레인 α-헬릭스 11 및 이들 사이의 서열은 SEQ ID NO : 8 을 포함한다) ; 및
- 재조합 폴리뉴클레오티드가 발현되는 배지에 세포를 배양하는 단계
(재조합 폴리뉴클레오티드가 포함되지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로 셀로덱스트린의 수송이 증가되었다).
- 제 1 항 내지 제 8 항 중 어느 한 항에 있어서,
상기 폴리펩티드는 NCU00801 또는 NCU08114 와 적어도 29 %, 적어도 30 %, 적어도 35 %, 적어도 40 %, 적어도 45 %, 적어도 50 %, 적어도 55 %, 적어도 60 %, 적어도 65 %, 적어도 70 %, 적어도 75 %, 적어도 80 %, 적어도 85 %, 적어도 90 %, 적어도 95 %, 적어도 99 % 또는 적어도 100 % 의 아미노산 동일성(amino acid identity)을 갖는 방법.
- 제 1 항 내지 제 9 항 중 어느 한 항에 있어서,
상기 숙주 세포는 β-글루코시다아제(β-glucosidase)의 적어도 촉매 도메인(catalytic domain)을 코딩하는 두 번째 재조합 폴리뉴클레오티드를 더 포함하는 방법.
- 제 10 항에 있어서,
상기 β-글루코시다아제는 네우로스포라 크라사(Neurospora crassa)로부터 온 것인 방법.
- 제 11 항에 있어서,
상기 β-글루코시다아제는 NCU00130 에 의해 코드되는 방법.
- 제 1 항 내지 제 12 항 중 어느 한 항에 있어서,
상기 숙주 세포는 하나 또는 그 이상의 재조합 폴리뉴클레오티드를 더 포함하고, 상기 하나 또는 그 이상의 폴리뉴클레오티드는 오탄당 이용(pentose utilization)에 포함되는 하나 또는 그 이상의 효소를 코드하는 방법.
- 제 13 항에 있어서,
상기 하나 또는 그 이상의 효소는, L-아라비노스 이성질화효소(L-arabinose isomerase), L-리블로인산화효소(L-ribulokinase), L-리블로스-5-P 4 에피머화효소(L- ribulose-5-P 4 epimerase), 자일로스 이성질화효소(xylose isomerase), 자일루로키나아제(xylulokinase), 알도스 환원효소(aldose reductase), L-아라비니톨 4-탈수소효소(L-arabinitol 4-dehydrogenase), L-자일루로스 환원효소(L-xylulose reductase) 및 자일리톨 탈수소효소로 이루어진 하나 또는 그 이상의 군으로부터 선택된 것인 방법.
- 제 1 항 내지 제 14 항 중 어느 한 항에 있어서,
상기 숙주 세포는 세 번째 재조합 폴리뉴클레오티드를 더 포함하고, 상기 세 번째 재조합 폴리뉴클레오티드는 오탄당 운반체(pentose transporter)를 코드하는 방법.
- 제 15 항에 있어서,
상기 오탄당 운반체는 NCU00821, NCU04963, NCU06138, STL12/XUT6, SUT2, SUT3, XUT1 및 XUT3 으로 이루어진 군으로부터 선택된 방법.
- 하기 단계를 포함하는, 세포의 성장을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 1 은 SEQ ID NO : 1 을 포함하고, 상기 폴리펩티드는 셀로덱스트린 운반체이다) ; 및
- 셀로덱스트린을 포함하는 배지에서 숙주 세포를 배양하는 단계
(상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 더 빠른 비율로 성장하였다).
- 하기 단계를 포함하는, 세포의 성장을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 2 는 SEQ ID NO : 2 를 포함하고, 상기 폴리펩티드는 셀로덱스트린 운반체이다) ; 및
- 셀로덱스트린을 포함하는 배지에서 숙주 세포를 배양하는 단계
(상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 더 빠른 비율로 성장하였다).
- 하기 단계를 포함하는, 세포의 성장을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 2 및 트랜스멤브레인 α-헬릭스 3 을 연결하는 루프는 SEQ ID NO : 3 을 포함하고, 상기 폴리펩티드는 셀로덱스트린 운반체이다) ; 및
- 셀로덱스트린을 포함하는 배지에서 숙주 세포를 배양하는 단계
(상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 더 빠른 비율로 성장하였다).
- 하기 단계를 포함하는, 세포의 성장을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 5 는 SEQ ID NO : 4 를 포함하고, 상기 폴리펩티드는 셀로덱스트린 운반체이다) ; 및
- 셀로덱스트린을 포함하는 배지에서 숙주 세포를 배양하는 단계
(상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 더 빠른 비율로 성장하였다).
- 하기 단계를 포함하는, 세포의 성장을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 6 은 SEQ ID NO : 5 를 포함하고, 상기 폴리펩티드는 셀로덱스트린 운반체이다) ; 및
- 셀로덱스트린을 포함하는 배지에서 숙주 세포를 배양하는 단계
(상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 더 빠른 비율로 성장하였다).
- 하기 단계를 포함하는, 세포의 성장을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 6 및 트랜스멤브레인 α-헬릭스 7 사이의 서열은 SEQ ID NO : 6 을 포함하고, 상기 폴리펩티드는 셀로덱스트린 운반체이다) ; 및
- 셀로덱스트린을 포함하는 배지에서 숙주 세포를 배양하는 단계
(상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 더 빠른 비율로 성장하였다).
- 하기 단계를 포함하는, 세포의 성장을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 7 은 SEQ ID NO : 7 을 포함하고, 상기 폴리펩티드는 셀로덱스트린 운반체이다) ; 및
- 셀로덱스트린을 포함하는 배지에서 숙주 세포를 배양하는 단계
(상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 더 빠른 비율로 성장하였다).
- 하기 단계를 포함하는, 세포의 성장을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 10 및 트랜스멤브레인 α-헬릭스 11 및 이들 사이의 서열은 SEQ ID NO : 8 을 포함하고, 상기 폴리펩티드는 셀로덱스트린 운반체이다) ; 및
- 셀로덱스트린을 포함하는 배지에서 숙주 세포를 배양하는 단계
(상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 배지에서 더 빠른 비율로 성장하였다).
- 제 17 항 내지 제 24 항 중 어느 한 항에 있어서,
상기 폴리펩티드는, NCU00801 또는 NCU08114 와 적어도 29 %, 적어도 30 %, 적어도 35 %, 적어도 40 %, 적어도 45 %, 적어도 50 %, 적어도 55 %, 적어도 60 %, 적어도 65 %, 적어도 70 %, 적어도 75 %, 적어도 80 %, 적어도 85 %, 적어도 90 %, 적어도 95 %, 적어도 99 % 또는 적어도 100 % 의 아미노산 동일성을 갖는 방법.
- 제 17 항 내지 제 25 항 중 어느 한 항에 있어서,
상기 숙주 세포는 내인성(endogenous) 또는 두 번째 재조합 폴리뉴클레오티드를 더 포함하고, 상기 폴리뉴클레오티드는 β-글루코시다아제의 적어도 촉매 도메인(catalytic domain)을 코드하는 방법.
- 제 26 항에 있어서,
상기 β-글루코시다아제는 네우로스포라 크라사(Neurospora crassa)로부터 온 것인 방법.
- 제 27 항에 있어서,
상기 β-글루코시다아제는 NCU00130 에 의해 코드되는 방법.
- 하기 단계를 포함하는, 셀룰로스-유도(cellulose-derived) 및 헤미셀룰로스-유도 당(hemicelluloses-derived sugars)을 공동 발효시키는(co-fermenting) 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 셀로덱스트린 운반체를 코딩하는 첫 번째 재조합 폴리뉴클레오티드 및 β-글루코시다아제의 촉매 도메인을 코딩하는 두 번째 재조합 폴리뉴클레오티드를 포함한다) ; 및
- 셀룰로스-유도 당 및 헤미셀룰로스-유도 당을 포함하는 배지에서 숙주 세포를 배양하는 단계
(상기 재조합 폴리뉴클레오티드의 발현은 셀룰로스-유도 당 및 헤미셀룰로스-유도 당을 공동 발효시킬 수 있다).
- 제 29 항에 있어서,
상기 첫 번째 재조합 폴리뉴클레오티드는, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하고, 트랜스멤브레인 α-헬릭스 1 은 SEQ ID NO : 1 을 포함하는 방법.
- 제 29 항에 있어서,
상기 첫 번째 재조합 폴리뉴클레오티드는, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하고, 트랜스멤브레인 α-헬릭스 2 는 SEQ ID NO : 2 를 포함하는 방법.
- 제 29 항에 있어서,
상기 첫 번째 재조합 폴리뉴클레오티드는, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하고, 트랜스멤브레인 α-헬릭스 2 및 트랜스멤브레인 α-헬릭스 3 을 연결하는 루프는 SEQ ID NO : 3 을 포함하는 방법.
- 제 29 항에 있어서,
상기 첫 번째 재조합 폴리뉴클레오티드는, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하고, 트랜스멤브레인 α-헬릭스 5 는 SEQ ID NO : 4 를 포함하는 방법.
- 제 29 항에 있어서,
상기 첫 번째 재조합 폴리뉴클레오티드는, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하고, 트랜스멤브레인 α-헬릭스 6 은 SEQ ID NO : 5 를 포함하는 방법.
- 제 29 항에 있어서,
상기 첫 번째 재조합 폴리뉴클레오티드는, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하고, 트랜스멤브레인 α-헬릭스 6 및 트랜스멤브레인 α-헬릭스 7 사이의 서열은 SEQ ID NO : 6 을 포함하는 방법.
- 제 29 항에 있어서,
상기 첫 번째 재조합 폴리뉴클레오티드는, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하고, 트랜스멤브레인 α-헬릭스 7 은 SEQ ID NO : 7 을 포함하는 방법.
- 제 29 항에 있어서,
상기 첫 번째 재조합 폴리뉴클레오티드는, 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코드하고, 트랜스멤브레인 α-헬릭스 10, 트랜스멤브레인 α-헬릭스 11 및 이들 사이의 서열은 SEQ ID NO : 8 을 포함하는 방법.
- 제 30 항 내지 제 37 항 중 어느 한 항에 있어서,
상기 폴리펩티드는 NCU00801 또는 NCU08114 와 적어도 29 %, 적어도 30 %, 적어도 35 %, 적어도 40 %, 적어도 45 %, 적어도 50 %, 적어도 55 %, 적어도 60 %, 적어도 65 %, 적어도 70 %, 적어도 75 %, 적어도 80 %, 적어도 85 %, 적어도 90 %, 적어도 95 %, 적어도 99 % 또는 적어도 100 % 의 아미노산 동일성을 갖는 방법.
- 제 29 항 내지 제 38 항 중 어느 한 항에 있어서,
상기 β-글루코시다아제는 네우로스포라 크라사(Neurospora crassa)로부터 온 것인 방법.
- 제 39 항에 있어서,
상기 β-글루코시다아제는 NCU00130 에 의해 코드되는 방법.
- 제 29 항 내지 제 40 항 중 어느 한 항에 있어서,
상기 숙주 세포는 하나 또는 그 이상의 재조합 폴리뉴클레오티드를 더 포함하고, 상기 하나 또는 그 이상의 폴리뉴클레오티드는 오탄당 이용(pentose utilization)에 포함되는 하나 또는 그 이상의 효소를 코드하는 방법.
- 제 41 항에 있어서,
상기 하나 또는 그 이상의 효소는, L-아라비노스 이성질화효소, L-리블로인산화효소, L-리블로스-5-P 4 에피머화효소, 자일로스 이성질화효소, 자일루로키나아제, 알도스 환원효소, L-아라비니톨 4-탈수소효소, L-자일루로스 환원효소 및 자일리톨 탈수소효소로 이루어진 하나 또는 그 이상의 군으로부터 선택된 것인 방법.
- 제 29 항 내지 제 42 항 중 어느 한 항에 있어서,
상기 숙주 세포는 세 번째 재조합 폴리뉴클레오티드를 더 포함하고, 상기 폴리뉴클레오티드는 오탄당 운반체(pentose transporter)를 코드하는 방법.
- 제 43 항에 있어서,
상기 오탄당 운반체는 NCU00821, NCU04963, NCU06138, STL12/XUT6, SUT2, SUT3, XUT1 및 XUT3 으로 이루어진 군으로부터 선택된 방법.
- 제 29 항 내지 제 44 항 중 어느 한 항에 있어서,
상기 셀룰로스-유도 당은 셀로비오스(cellobiose), 셀로트리오스(cellotriose) 및 셀로테트라오스(celltetraose)로 이루어진 군으로부터 선택된 것이고, 헤미셀룰로스-유도 당은 자일로스인 방법.
- 하기 단계를 포함하는, 숙주 세포에 의한 탄화수소(hydrocarbon) 또는 탄화수소 유도체(hydrocarbon derivatives)의 합성을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 1 은 SEQ ID NO : 1 을 포함하고, 상기 폴리펩티드는 탄화수소 또는 탄화수소 유도체를 합성하기 위해 숙주 세포 내의 셀로덱스트린을 수송한다) ; 및
- 숙주 세포에 의해 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 셀로덱스트린 또는 셀로덱스트린의 근원(source)을 포함하는 배지에서 숙주 세포를 배양하는 단계
(상기 세포 내로 셀로덱스트린의 수송은 재조합 폴리뉴클레오티드의 발현 상에서 증가되었다).
- 하기 단계를 포함하는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 2 는 SEQ ID NO : 2 를 포함하고, 상기 폴리펩티드는 탄화수소 또는 탄화수소 유도체를 합성하기 위해 숙주 세포 내로 셀로덱스트린을 수송한다) ; 및
- 숙주 세포에 의해 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 셀로덱스트린 또는 셀로덱스트린의 근원을 포함하는 배지에서 숙주 세포를 배양하는 단계
(상기 세포 내로 셀로덱스트린의 수송은 재조합 폴리뉴클레오티드의 발현 상에서 증가되었다).
- 하기 단계를 포함하는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 2 및 트랜스멤브레인 α-헬릭스 3 을 연결하는 루프는 SEQ ID NO : 3 을 포함하고, 상기 폴리펩티드는 탄화수소 또는 탄화수소 유도체를 합성하기 위해 숙주 세포 내로 셀로덱스트린을 수송한다) ; 및
- 숙주 세포에 의해 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 셀로덱스트린 또는 셀로덱스트린의 근원을 포함하는 배지에서 숙주 세포를 배양하는 단계
(상기 세포 내로 셀로덱스트린의 수송은 재조합 폴리뉴클레오티드의 발현 상에서 증가되었다).
- 하기 단계를 포함하는, 숙주 세포에 의해 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 5 는 SEQ ID NO : 4 를 포함하고, 상기 폴리펩티드는 탄화수소 또는 탄화수소 유도체를 합성하기 위해 숙주 세포 내로 셀로덱스트린을 수송한다) ; 및
- 숙주 세포에 의해 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 셀로덱스트린 또는 셀로덱스트린의 근원을 포함하는 배지에서 숙주 세포를 배양하는 단계
(상기 세포 내로 셀로덱스트린의 수송은 재조합 폴리뉴클레오티드의 발현 상에서 증가되었다).
- 하기 단계를 포함하는, 숙주 세포에 의해 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 6 은 SEQ ID NO : 5 를 포함하고, 상기 폴리펩티드는 탄화수소 또는 탄화수소 유도체를 합성하기 위해 숙주 세포 내로 셀로덱스트린을 수송한다) ; 및
- 숙주 세포에 의해 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 셀로덱스트린 또는 셀로덱스트린의 근원을 포함하는 배지에서 숙주 세포를 배양하는 단계
(상기 세포 내로 셀로덱스트린의 수송은 재조합 폴리뉴클레오티드의 발현 상에서 증가되었다).
- 하기 단계를 포함하는, 숙주 세포에 의해 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 6 및 트랜스멤브레인 α-헬릭스 7 사이의 서열은 SEQ ID NO : 6 을 포함하고, 상기 폴리펩티드는 탄화수소 또는 탄화수소 유도체를 합성하기 위해 숙주 세포 내로 셀로덱스트린을 수송한다) ; 및
- 숙주 세포에 의해 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 셀로덱스트린 또는 셀로덱스트린의 근원을 포함하는 배지에서 숙주 세포를 배양하는 단계
(상기 세포 내로 셀로덱스트린의 수송은 재조합 폴리뉴클레오티드의 발현 상에서 증가되었다).
- 하기 단계를 포함하는, 숙주 세포에 의해 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 7 은 SEQ ID NO : 7 을 포함하고, 상기 폴리펩티드는 탄화수소 또는 탄화수소 유도체를 합성하기 위해 숙주 세포 내로 셀로덱스트린을 수송한다) ; 및
- 숙주 세포에 의해 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 셀로덱스트린 또는 셀로덱스트린의 근원을 포함하는 배지에서 숙주 세포를 배양하는 단계
(상기 세포 내로 셀로덱스트린의 수송은 재조합 폴리뉴클레오티드의 발현 상에서 증가되었다).
- 하기 단계를 포함하는, 숙주 세포에 의해 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 트랜스멤브레인 α-헬릭스 1, α-헬릭스 2, α-헬릭스 3, α-헬릭스 4, α-헬릭스 5, α-헬릭스 6, α-헬릭스 7, α-헬릭스 8, α-헬릭스 9, α-헬릭스 10, α-헬릭스 11, α-헬릭스 12 를 포함하는 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 트랜스멤브레인 α-헬릭스 10 및 트랜스멤브레인 α-헬릭스 11 및 이들 사이의 서열은 SEQ ID NO : 8 을 포함하고, 상기 폴리펩티드는 탄화수소 또는 탄화수소 유도체를 합성하기 위해 숙주 세포 내로 셀로덱스트린을 수송한다) ; 및
- 숙주 세포에 의해 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 셀로덱스트린 또는 셀로덱스트린의 근원을 포함하는 배지에서 숙주 세포를 배양하는 단계
(상기 세포 내로의 셀로덱스트린의 수송은 재조합 폴리뉴클레오티드의 발현 상에서 증가되었다).
- 제 46 항 내지 제 53 항 중 어느 한 항에 있어서,
상기 폴리펩티드는, NCU00801 또는 NCU08114 와 적어도 29 %, 적어도 30 %, 적어도 35 %, 적어도 40 %, 적어도 45 %, 적어도 50 %, 적어도 55 %, 적어도 60 %, 적어도 65 %, 적어도 70 %, 적어도 75 %, 적어도 80 %, 적어도 85 %, 적어도 90 %, 적어도 95 %, 적어도 99 % 또는 적어도 100 % 의 아미노산 동일성을 갖는 방법.
- 제 46 항 내지 제 54 항 중 어느 한 항에 있어서,
상기 숙주 세포는 두 번째 재조합 폴리뉴클레오티드를 더 포함하고, 상기 폴리뉴클레오티드는 β-글루코시다아제(β-glucosidase)의 적어도 촉매 도메인을 코드하는 방법.
- 제 55 항에 있어서,
상기 β-글루코시다아제는 네우로스포라 크라사(Neurospora crassa)로부터 온 것인 방법.
- 제 56 항에 있어서,
상기 β-글루코시다아제는 NCU00130 에 의해 코드되는 방법.
- 제 46 항 내지 제 57 항 중 어느 한 항에 있어서,
상기 셀로덱스트린의 근원은 셀룰로스를 포함하는 방법.
- 제 46 항 내지 제 58 항 중 어느 한 항에 있어서,
상기 탄화수소 또는 탄화수소 유도체는 연료(fuel)로서 사용될 수 있는 방법.
- 제 59 항에 있어서,
상기 탄화수소 또는 탄화수소 유도체는 에탄올을 포함하는 방법.
- 제 59 항에 있어서,
상기 탄화수소 또는 탄화수소 유도체는 부탄올을 포함하는 방법.
- 제 1 항 내지 제 61 항 중 어느 한 항에 있어서,
상기 배지는 변경된 유기체(altered organism)로부터 셀룰라아제-함유 효소 혼합물을 포함하고, 변경되지 않은 유기체(unaltered organism)로부터 셀룰라아제-함유 혼합물과 비교하여, 상기 셀룰라아제-함유 혼합물은 감소된 β-글루코시다아제 활성도(β-glucosidase activity)를 갖는 방법.
- 제 1 항 내지 제 62 항 중 어느 한 항에 있어서,
상기 숙주 세포는, 사카로마이시스 ( Saccharomyces sp .), 사카로미세스 세레비시아( Saccharomyces cerevisiae ), 사카로미세스 모나센시스( Saccharomyces monacensis), 사카로미세스 바야누스 ( Saccharomyces bayanus ), 사카로미세스 파스토리아누스( Saccharomyces pastorianus ), 사카로미세스 칼스베르겐시스( Saccharomyces carlsbergensis ), 사카로미세스 폼베 ( Saccharomyces pombe ), 클루이베르마이세스( Kluyveromyces sp .), 클루이베르마이세스 마르시암우스( Kluyveromyces marxiamus ), 클루이베르마이세스 락티스( Kluyveromyces lactis), 클루이베르마이세스 프라길리스 ( Kluyveromyces fragilis ), 피치아 스티피 티스( Pichia stipitis ), 스포로트리쿰 서머파일 ( Sporotrichum thermophile ), 칸디다 쉬하테( Candida shehatae ), 칸디다 트로피칼리스 ( Candida tropicalis ), 네우로스포라 크라사 ( Neurospora crassa ), 자이모나스 모빌리스( Zymomonas mobilis ), 클로스트리듐( Clostridium sp .), 클로스트리듐 파이토퍼멘탄스( Clostridium phytofermentans), 클로스트리듐 써모셀럼 ( Clostridium thermocellum ), 클로스트리듐 베이저린키이( Clostridium beijerinckii ), 클로스트리듐 아세토부틸리쿰( Clostridium acetobutylicum ), 무렐라 서모아세티카 ( Moorella thermoacetica ), 대장균( Escherichia coli ), 크레브시엘라 옥시토카 ( Klebsielle oxytoca ), 서모안아에로박테리움 사카로락티쿰 ( Thermoanaerobacterium saccharolyticum ) 및 고초균( Bacillus subtilis )으로 이루어진 군으로부터 선택한 방법.
- 제 1 항 내지 제 63 항 어느 한 항에 있어서,
상기 셀로덱스트린은, 셀로비오스(cellobiose), 셀로트리오스(cellotriose) 및 셀로테트라오스(cellotetraose)로 이루어진 하나 또는 그 이상의 군으로부터 선택된 방법.
- NCU00821 및 STL12/XUT6 로 이루어진 군으로부터 선택한 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포로서, 상기 폴리펩티드는 세포 내로 자일로스를 수송하는 숙주 세포.
- XUT1 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포로서, 상기 폴리펩티드는 세포 내로 아라비노스(arabinose)를 수송하는 숙주 세포.
- NCU06138 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포로서, 상기 폴리펩티드는 세포 내로 아라비노스 및 글루코스를 수송하는 숙주 세포.
- SUT2, SUT3 및 XUT3 으로 이루어진 군으로부터 선택된 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포로서, 상기 폴리펩티드는 세포 내로 자일로스 및 글루코스를 수송하는 숙주 세포.
- NCU04963 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하는 숙주 세포로서, 상기 폴리펩티드는 세포 내로 자일로스, 아라비노스 및 글루코스를 수송하는 숙주 세포.
- 제 65 항 내지 제 69 항 중 어느 한 항에 있어서,
하나 또는 그 이상의 재조합 폴리뉴클레오티드를 더 포함하고, 상기 하나 또는 그 이상의 폴리뉴클레오티드는 오탄당 이용에 포함되는 하나 또는 그 이상의 효소를 코드하는 숙주 세포.
- 제 70 항에 있어서,
상기 하나 또는 그 이상의 효소는, L-아라비노스 이성질화효소, L-리블로인산화효소, L-리블로스-5-P 4 에피머화효소, 자일로스 이성질화효소, 자일루로키나아제, 알도스 환원효소, L-아라비니톨 4-탈수소효소, L-자일루로스 환원효소 및 자일리톨 탈수소효소로 이루어진 하나 또는 그 이상의 군으로부터 선택된 것인 방법.
- 제 65 항 내지 제 71 항 중 어느 한 항에 있어서,
상기 숙주 세포는, 사카로마이시스 ( Saccharomyces sp .), 사카로미세스 세레비시아( Saccharomyces cerevisiae ), 사카로미세스 모나센시스( Saccharomyces monacensis), 사카로미세스 바야누스 ( Saccharomyces bayanus ), 사카로미세스 파스토리아누스( Saccharomyces pastorianus ), 사카로미세스 칼스베르겐시스( Saccharomyces carlsbergensis ), 사카로미세스 폼베 ( Saccharomyces pombe ), 클루이베르마이세스( Kluyveromyces sp .), 클루이베르마이세스 마르시암우스( Kluyveromyces marxiamus ), 클루이베르마이세스 락티스( Kluyveromyces lactis), 클루이베르마이세스 프라길리스 ( Kluyveromyces fragilis ), 피치아 스티피티스( Pichia stipitis ), 스포로트리쿰 서머파일 ( Sporotrichum thermophile ), 칸디다 쉬하테( Candida shehatae ), 칸디다 트로피칼리스 ( Candida tropicalis ), 네우로스포라 크라사 ( Neurospora crassa ), 자이모나스 모빌리스( Zymomonas mobilis ), 클로스트리듐( Clostridium sp .), 클로스트리듐 파이토퍼멘탄스( Clostridium phytofermentans), 클로스트리듐 써모셀럼 ( Clostridium thermocellum ), 클로스트리듐 베이저린키이( Clostridium beijerinckii ), 클로스트리듐 아세토부틸리쿰( Clostridium acetobutylicum ), 무렐라 서모아세티카 ( Moorella thermoacetica ), 대장균( Escherichia coli ), 크레브시엘라 옥시토카 ( Klebsielle oxytoca ), 서모안아에로박테리움 사카로락티쿰 ( Thermoanaerobacterium saccharolyticum ) 및 고초균( Bacillus subtilis )으로 이루어진 군으로부터 선택된 방법.
- 하기 단계를 포함하는, 세포 내로 자일로스의 수송을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는, NCU00821 및 STL12/XUT6 로 이루어진 군으로부터 선택된 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함한다) ; 및
- 재조합 폴리뉴클레오티드가 발현되는 세포를 배양하는 단계
(재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로 자일로스의 수송이 증가되었다).
- 하기 단계를 포함하는, 세포 내로 아라비노스의 수송을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 XUT1 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함한다) ; 및
- 재조합 폴리뉴클레오티드가 발현되는 세포를 배양하는 단계
(재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로 아라비노스의 수송이 증가되었다).
- 하기 단계를 포함하는, 세포 내로 아라비노스 또는 글루코스의 수송을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 NCU06138 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함한다) ; 및
- 재조합 폴리뉴클레오티드가 발현되는 세포를 배양하는 단계
(재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로 아라비노스 또는 글루코스의 수송이 증가되었다).
- 하기 단계를 포함하는, 세포 내로 자일로스 또는 글루코스의 수송을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 SUT2, SUT3 및 XUT3 으로 이루어진 군으로부터 선택된 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함한다) ; 및
- 재조합 폴리뉴클레오티드가 발현되는 세포를 배양하는 단계
(재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로 자일로스 또는 글루코스의 수송이 증가되었다).
- 하기 단계를 포함하는, 세포 내로 자일로스, 아라비노스 또는 글루코스의 수송을 증가시키는 방법 :
- 숙주 세포를 준비하는 단계
(상기 숙주 세포는 NCU04963 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함한다) ; 및
- 재조합 폴리뉴클레오티드가 발현되는 세포를 배양하는 단계
(재조합 폴리뉴클레오티드를 포함하지 않는 세포와 비교하여, 상기 재조합 폴리뉴클레오티드의 발현으로 세포 내로 자일로스, 아라비노스 또는 글루코스의 수송이 증가되었다).
- 제 73 항 내지 제 77 항 중 어느 한 항에 있어서,
하나 또는 그 이상의 재조합 폴리뉴클레오티드를 더 포함하고, 상기 하나 또는 그 이상의 폴리뉴클레오티드는 오탄당 이용에서 포함되는 하나 또는 그 이상의 효소를 코드하는 방법.
- 제 78 항에 있어서,
상기 하나 또는 그 이상의 효소는 L-아라비노스 이성질화효소, L-리블로인산화효소, L-리블로스-5-P 4 에피머화효소, 자일로스 이성질화효소, 자일루로키나아제, 알도스 환원효소, L-아라비니톨 4-탈수소효소, L-자일루로스 환원효소 및 자일리톨 탈수소효소로 이루어진 하나 또는 그 이상 군으로부터 선택된 것인 방법.
- 하기 단계를 포함하는, 세포의 성장을 증가시키는 방법 :
- 숙주 세포를 제공하는 단계
(상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리뉴클레오티드는 NCU00821 및 STL12/XUT6 로 이루어진 군으로부터 선택된 폴리펩티드를 코드하고, 상기 폴리펩티드는 자일로스를 수송한다) ; 및
- 자일로스를 포함하는 배지에서 숙주 세포를 배양하는 단계
(재조합 폴리뉴클레오티드가 포함되지 않는 세포와 비교하여, 상기 숙주 세포는 배지에서 더 빠른 비율로 성장하였다).
- 하기 단계를 포함하는, 세포의 성장을 증가시키는 방법 :
- 숙주 세포를 제공하는 단계
(상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리뉴클레오티드는 XUT1 폴리펩티드를 코드하고, 상기 폴리펩티드는 아라비노스를 수송한다) ; 및
- 아라비노스를 포함하는 배지에서 숙주 세포를 배양하는 단계
(재조합 폴리뉴클레오티드가 포함되지 않는 세포와 비교하여, 상기 숙주 세포는 배지에서 더 빠른 비율로 성장하였다).
- 하기 단계를 포함하는, 세포의 성장을 증가시키는 방법 :
- 숙주 세포를 제공하는 단계
(상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리뉴클레오티드는 NCU06138 폴리펩티드를 코드하고, 상기 폴리펩티드는 아라비노스 및 글루코스를 수송한다) ; 및
- 아라비노스 또는 글루코스를 포함하는 배지에서 숙주 세포를 배양하는 단계
(재조합 폴리뉴클레오티드가 포함되지 않는 세포와 비교하여, 상기 숙주 세포는 배지에서 더 빠른 비율로 성장하였다).
- 하기 단계를 포함하는, 세포의 성장을 증가시키는 방법 :
- 숙주 세포를 제공하는 단계
(상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리뉴클레오티드는 SUT2, SUT3 및 XUT3 으로 이루어진 군으로부터 선택된 폴리펩티드를 코드하고, 상기 폴리펩티드는 자일로스 및 글루코스를 수송한다) ; 및
- 자일노스 또는 글루코스를 포함하는 배지에서 숙주 세포를 배양하는 단계
(재조합 폴리뉴클레오티드가 포함되지 않는 세포와 비교하여, 상기 숙주 세포는 배지에서 더 빠른 비율로 성장하였다).
- 하기 단계를 포함하는, 세포의 성장을 증가시키는 방법 :
- 숙주 세포를 제공하는 단계
(상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리뉴클레오티드는 NCU04963 폴리펩티드를 코드하고, 상기 폴리펩티드는 자일로스, 아라비노스 및 글루코스를 수송한다) ; 및
- 자일노스, 아라비노스 또는 글루코스를 포함하는 배지에서 숙주 세포를 배양하는 단계
(재조합 폴리뉴클레오티드가 포함되지 않는 세포와 비교하여, 상기 숙주 세포는 배지에서 더 빠른 비율로 성장하였다).
- 제 80 항 내지 제 84 항 중 어느 한 항에 있어서,
상기 숙주 세포는, 오탄당 이용에 포함되는 하나 또는 그 이상의 효소를 코딩하는 하나 또는 그 이상의 내인성(endogenous) 또는 재조합 폴리뉴클레오티드를 더 포함하는 방법.
- 제 85 항에 있어서,
상기 하나 또는 그 이상의 효소는, L-아라비노스 이성질화효소, L-리블로인산화효소, L-리블로스-5-P 4 에피머화효소, 자일로스 이성질화효소, 자일루로키나아제, 알도스 환원효소, L-아라비니톨 4-탈수소효소, L-자일루로스 환원효소 및 자일리톨 탈수소효소로 이루어진 하나 또는 그 이상의 군으로부터 선택된 것인 방법.
- 하기 단계를 포함하는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체(hydrocarbon derivatives)의 합성을 증가시키는 방법 :
- 숙주 세포를 제공하는 단계
(상기 숙주 세포는, NCU00821 및 STL12/XUT6 로 이루어진 군으로부터 선택된 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리펩티드는 탄화수소 또는 탄화수소 유도체를 합성하기 위해 숙주 세포 내로 자일로스를 수송한다) ; 및
- 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 자일로스 또는 자일로스 근원을 포함하는 배지에 숙주 세포를 배양하는 단계
(세포 내로 자일로스 수송은 재조합 폴리뉴클레오티드의 발현 상에서 증가되었다).
- 하기 단계를 포함하는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법 :
- 숙주 세포를 제공하는 단계
(상기 숙주 세포는, XUTl 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리펩티드는 탄화수소 또는 탄화수소 유도체를 합성하기 위해 숙주 세포 내로 아라비노스를 수송한다) ; 및
- 숙주 세포에 의해 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 아라비노스 또는 아라비노스 근원을 포함하는 배지에 숙주 세포를 배양하는 단계
(세포 내로 아라비노스 수송은 재조합 폴리뉴클레오티드의 발현 상에서 증가되었다).
- 하기 단계를 포함하는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법 :
- 숙주 세포를 제공하는 단계
(상기 숙주 세포는 NCU06138 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리펩티드는 탄화수소 또는 탄화수소 유도체를 합성하기 위해 숙주 세포 내로 아라비노스 또는 글루코스를 수송한다) ; 및
- 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 아라비노스, 글루코스, 아라비노스 근원 또는 글루코스 근원을 포함하는 배지에 숙주 세포를 배양하는 단계
(세포 내로 아라비노스 또는 글루코스 수송은 재조합 폴리뉴클레오티드의 발현 상에서 증가되었다).
- 하기 단계를 포함하는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법 :
- 숙주 세포를 제공하는 단계
(상기 숙주 세포는, SUT2, SUT3, 및 XUT3 으로 이루어진 군으로부터 선택된 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리펩티드는 탄화수소 또는 탄화수소 유도체를 합성하기 위해 숙주 세포 내로 자일로스 또는 글루코스를 수송한다) ; 및
- 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 자일로스, 글루코스, 자일로스 근원 또는 글루코스 근원을 포함하는 배지에 숙주 세포를 배양하는 단계
(세포 내로 자일로스 또는 글루코스 수송은 재조합 폴리뉴클레오티드의 발현 상에서 증가되었다).
- 하기 단계를 포함하는, 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키는 방법 :
- 숙주 세포를 제공하는 단계
(상기 숙주 세포는, NCU04963 폴리펩티드를 코딩하는 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리펩티드는 탄화수소 또는 탄화수소 유도체를 합성하기 위해 숙주 세포 내로 자일로스, 아라비노스 또는 글루코스를 수송한다) ; 및
- 숙주 세포에 의한 탄화수소 또는 탄화수소 유도체의 합성을 증가시키기 위해 자일로스, 아라비노스, 글루코스, 자일로스 근원, 아라비노스 근원 또는 글루코스 근원을 포함하는 배지에 숙주 세포를 배양하는 단계
(세포 내로 자일로스, 아라비노스 또는 글루코스 수송은 재조합 폴리뉴클레오티드의 발현 상에서 증가되었다).
- 제 89 항 내지 제 91 항 중 어느 한 항에 있어서,
상기 글루코스의 근원은 셀룰로스를 포함하는 방법.
- 제 87 항 내지 제 92 항 중 어느 한 항에 있어서,
자일로스 또는 아라비노스의 근원은 헤미셀룰로스를 포함하는 방법.
- 제 87 항 내지 제 93 항 중 어느 한 항에 있어서,
상기 탄화수소 또는 탄화수소 유도체는 연료(fuel)로서 사용될 수 있는 방법.
- 제 94 항에 있어서,
상기 탄화수소 또는 탄화수소 유도체는 에탄올을 포함하는 방법.
- 제 81 항에 있어서,
상기 탄화수소 또는 탄화수소 유도체는 부탄올을 포함하는 방법.
- 제 73 항 내지 제 95 항 중 어느 한 항에 있어서,
상기 숙주 세포는, 사카로마이시스 ( Saccharomyces sp .), 사카로미세스 세레비시아( Saccharomyces cerevisiae ), 사카로미세스 모나센시스( Saccharomyces monacensis), 사카로미세스 바야누스 ( Saccharomyces bayanus ), 사카로미세스 파스토리아누스( Saccharomyces pastorianus ), 사카로미세스 칼스베르겐시스( Saccharomyces carlsbergensis ), 사카로미세스 폼베 ( Saccharomyces pombe ), 클루이베르마이세스( Kluyveromyces sp .), 클루이베르마이세스 마르시암우 스( Kluyveromyces marxiamus ), 클루이베르마이세스 락티스( Kluyveromyces lactis), 클루이베르마이세스 프라길리스 ( Kluyveromyces fragilis ), 피치아 스티피티스( Pichia stipitis ), 스포로트리쿰 서머파일 ( Sporotrichum thermophile ), 칸디다 쉬하테( Candida shehatae ), 칸디다 트웨피칼리스 ( Candida twpicalis ), 네우로스포라 크라사 ( Neurospora crassa ), 자이모나스 모빌리스( Zymomonas mobilis ), 클로스트리듐( Clostridium sp .), 클로스트리듐 파이토퍼멘탄스( Clostridium phytofermentans), 클로스트리듐 써모셀럼 ( Clostridium thermocellum ), 클로스트리듐 베이저린키이( Clostridium beijerinckii ), 클로스트리듐 아세토부틸리쿰( Clostridium acetobutylicum ), 무렐라 서모아세티카 ( Moorella thermoacetica ), 대장균( Escherichia coli ), 크레브시엘라 옥시토카 ( Klebsielle oxytoca ), 서모안아에로박테리움 사카웰락티쿰 ( Thermoanaerobacterium sacchawlyticum ) 및 고초균( Bacillus subtilis )으로 이루어진 군으로부터 선택된 방법.
- 하기 단계를 포함하는, 세포의 성장을 증가시키는 방법 :
- 숙주 세포를 제공하는 단계
(상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리뉴클레오티드는 NCU07705 폴리펩티드를 코드한다) ; 및
- 셀룰로스를 포함하는 배지에 세포를 배양하는 단계
(재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 상기 숙주 세포는 배지에서 더 빠른 비율로 성장하였다).
- 제 98 항에 있어서,
상기 숙주 세포는, 사카로마이시스 ( Saccharomyces sp .), 사카로미세스 세레비시아( Saccharomyces cerevisiae ), 사카로미세스 모나센시스( Saccharomyces monacensis), 사카로미세스 바야누스 ( Saccharomyces bayanus ), 사카로미세스 파스토리아누스( Saccharomyces pastorianus ), 사카로미세스 칼스베르겐시스( Saccharomyces carlsbergensis ), 사카로미세스 폼베 ( Saccharomyces pombe ), 클루이베르마이세스( Kluyveromyces sp .), 클루이베르마이세스 마르시암우스( Kluyveromyces marxiamus ), 클루이베르마이세스 락티스( Kluyveromyces lactis), 클루이베르마이세스 프라길리스 ( Kluyveromyces fragilis ), 피치아 스티피 티스( Pichia stipitis ), 스포로트리쿰 서머파일 ( Sporotrichum thermophile ), 칸디 다 쉬하테( Candida shehatae ), 칸디다 트웨피칼리스 ( Candida twpicalis ), 네우로스포라 크라사 ( Neurospora crassa ), 자이모나스 모빌리스( Zymomonas mobilis ), 클로스트리듐( Clostridium sp .), 클로스트리듐 파이토퍼멘탄스( Clostridium phytofermentans), 클로스트리듐 써모셀럼 ( Clostridium thermocellum ), 클로스트리듐 베이저린키이( Clostridium beijerinckii ), 클로스트리듐 아세토부틸리쿰( Clostridium acetobutylicum ), 무렐라 서모아세티카 ( Moorella thermoacetica ), 대장균( Escherichia coli ), 크레브시엘라 옥시토카 ( Klebsielle oxytoca ), 서모안아에로박테리움 사카웰락티쿰 ( Thermoanaerobacterium sacchawlyticum ) 및 고초균( Bacillus subtilis )으로 이루어진 군으로부터 선택된 방법.
- 제 98 항에 있어서,
상기 숙주 세포는, 재조합 폴리뉴클레오티드에 작동가능하게 연결된(operably linked) 유도성 프로모터(inducible promoter)를 더 포함하는 방법.
- 제 98 항에 있어서,
셀룰라아제(cellulases)의 발현은 재조합 폴리뉴클레오티드의 발현 상에서 숙주 세포에서 증가되는 방법.
- 하기 단계를 포함하는, 바이오매스 중합체(biomass polymer)에서 세포의 성장을 증가시키는 방법 :
- 숙주 세포를 제공하는 단계
(상기 숙주 세포는 내인성 폴리뉴클레오티드를 포함하고, 상기 폴리뉴클레오티드는 NCU05137 폴리펩티드를 코드한다) ;
- 내인성 폴리뉴클레오티드의 발현을 저해하는 단계 ; 및
- 바이오매스 중합체를 포함하는 배지에서 세포를 배양하는 단계
(내인성 폴리뉴클레오티드의 발현을 저해하지 않는 세포보다, 상기 숙주 세포는 배지에서 더 빠른 비율로 성장하였다).
- 제 102 항에 있어서,
상기 숙주 세포는, 사카로마이시스 ( Saccharomyces sp .), 사카로미세스 세레비시아( Saccharomyces cerevisiae ), 사카로미세스 모나센시스( Saccharomyces monacensis), 사카로미세스 바야누스 ( Saccharomyces bayanus ), 사카로미세스 파스토리아누스( Saccharomyces pastorianus ), 사카로미세스 칼스베르겐시스( Saccharomyces carlsbergensis ), 사카로미세스 폼베 ( Saccharomyces pombe ), 클루이베르마이세스( Kluyveromyces sp .), 클루이베르마이세스 마르시암우스( Kluyveromyces marxiamus ), 클루이베르마이세스 락티스( Kluyveromyces lactis), 클루이베르마이세스 프라길리스 ( Kluyveromyces fragilis ), 피치아 스티피티스( Pichia stipitis ), 스포로트리쿰 서머파일 ( Sporotrichum thermophile ), 칸디다 쉬하테( Candida shehatae ), 칸디다 트웨피칼리스 ( Candida twpicalis ), 네우로스포라 크라사 ( Neurospora crassa ), 자이모나스 모빌리스( Zymomonas mobilis ), 클로 스트리듐( Clostridium sp .), 클로스트리듐 파이토퍼멘탄스( Clostridium phytofermentans), 클로스트리듐 써모셀럼 ( Clostridium thermocellum ), 클로스트리듐 베이저린키이( Clostridium beijerinckii ), 클로스트리듐 아세토부틸리쿰( Clostridium acetobutylicum ), 무렐라 서모아세티카 ( Moorella thermoacetica ), 대장균( Escherichia coli ), 크레브시엘라 옥시토카 ( Klebsielle oxytoca ), 서모안아에로박테리움 사카로락티쿰 ( Thermoanaerobacterium saccharolyticum ) 및 고초균( Bacillus subtilis )으로 이루어진 군으로부터 선택된 방법.
- 제 102 항에 있어서,
상기 숙주 세포의 셀룰라아제 활성도(cellulose activity)는 내인성 폴리뉴클레오티드 발현의 저해(inhibiting) 상에서 증가되는 방법.
- 제 102 항에 있어서,
상기 숙주 세포의 헤미셀룰라아제 활성도(hemicellulase activity)는 내인성 폴리뉴클레오티드 발현의 저해 상에서 증가되는 방법.
- 제 102 항에 있어서,
내인성 폴리뉴클레오티드 발현의 저해는 내인성 폴리뉴클레오티드를 포함하는 유전자(gene)의 돌연변이(mutating) 또는 결실(deleting)을 포함하는 방법.
- 제 102 항에 있어서,
상기 바이오매스 중합체는 셀룰로스인 방법.
- 제 102 항에 있어서,
상기 바이오매스 중합체는 헤미셀룰로스인 방법.
- 하기 단계를 포함하는, 세포의 성장을 증가시키는 방법 :
- 숙주 세포를 제공하는 단계
(상기 숙주 세포는 재조합 폴리뉴클레오티드를 포함하고, 상기 폴리뉴클레오티드는 NCU01517, NCU09133 및 NCU10040 으로 이루어진 군으로부터 선택된 폴리펩티드를 코드한다) ; 및
- 헤미셀룰로스를 포함하는 배지에 세포를 배양하는 단계
(재조합 폴리뉴클레오티드를 포함하지 않는 세포보다, 상기 숙주 세포는 배지에서 더 빠른 비율로 성장하였다).
- 제 109 항에 있어서,
상기 숙주 세포는, 사카로마이시스 ( Saccharomyces sp .), 사카로미세스 세레비시아( Saccharomyces cerevisiae ), 사카로미세스 모나센시스( Saccharomyces monacensis), 사카로미세스 바야누스 ( Saccharomyces bayanus ), 사카로미세스 파스토리아누스( Saccharomyces pastorianus ), 사카로미세스 칼스베르겐시스( Saccharomyces carlsbergensis ), 사카로미세스 폼베 ( Saccharomyces pombe ), 클루이베르마이세스( Kluyveromyces sp .), 클루이베르마이세스 마르시암우스( Kluyveromyces marxiamus ), 클루이베르마이세스 락티스( Kluyveromyces lactis), 클루이베르마이세스 프라길리스 ( Kluyveromyces fragilis ), 피치아 스티피티스( Pichia stipitis ), 스포로트리쿰 서머파일 ( Sporotrichum thermophile ), 칸디다 쉬하테( Candida shehatae ), 칸디다 트웨피칼리스 ( Candida twpicalis ), 네우로스포라 크라사 ( Neurospora crassa ), 자이모나스 모빌리스( Zymomonas mobilis ), 클로 스트리듐( Clostridium sp .), 클로스트리듐 파이토퍼멘탄스( Clostridium phytofermentans), 클로스트리듐 써모셀럼 ( Clostridium thermocellum ), 클로스트리듐 베이저린키이( Clostridium beijerinckii ), 클로스트리듐 아세토부틸리쿰( Clostridium acetobutylicum ), 무렐라 서모아세티카 ( Moorella thermoacetica ), 대장균( Escherichia coli ), 크레브시엘라 옥시토카 ( Klebsielle oxytoca ), 서모안아에로박테리움 사카로락티쿰 ( Thermoanaerobacterium saccharolyticum ) 및 고초균( Bacillus subtilis )으로 이루어진 군으로부터 선택된 방법.
- 제 109 항에 있어서,
상기 숙주 세포는, 재조합 폴리뉴클레오티드에 작동가능하게 연결된 유도성 프로모터(inducible promoter)를 더 포함하는 방법.
- 제 109 항에 있어서,
상기 숙주 세포의 헤미셀룰라아제 활성도는 재조합 폴리뉴클레오티드의 발현 상에서 증가되는 방법.
- 하기 단계를 포함하는, 셀룰로스를 분해하는(degrading) 방법 :
- 셀룰로스를 포함하는 조성물(composition)을 제공하는 단계 ; 및
- 변경된 유기체로부터 셀룰라아제-함유 효소 혼합물(cellulase-containing enzyme mixture)을 상기 조성물과 접촉시키는 단계
(변경되지 않는 유기체로부터 셀룰라아제-함유 혼합물을 비교하여, 상기 셀룰라아제-함유 혼합물은 감소된 β-글루코시다아제 활성도를 갖고, 상기 셀룰로스는 셀룰라아제-함유 혼합물에 의해 분해되었다).
- 제 113 항에 있어서,
상기 유기체는 β-글루코시다아제를 코딩하는 유전자의 돌연변이(mutation)에 의해 변형되는(altered) 방법.
- 제 113 항에 있어서,
상기 유기체는 β-글루코시다아제 발현의 감소에 의해 변형되는 방법.
- 제 113 항 내지 제 115 항 중 어느 한 항에 있어서,
상기 유기체는 균류(fungus) 및 세균(bacterium)으로 이루어진 군으로부터 선택된 방법.
- 제 116 항에 있어서,
상기 유기체는 사상균(filamentous fungus)인 방법.
- 제 113 항 내지 제 117 항 중 어느 한 항에 있어서,
상기 셀룰로스는 식물 물질(plant material)로부터 온 것인 방법.
- 제 118 항에 있어서,
상기 식물 물질은, 지팽이풀(switchgrass), 미스칸터스 ( Miscanthus ), 왕겨(rice hull), 버개스(bagasse), 아마(flax), 대나무(bamboo), 사이잘(sisal), 마닐라삼섬유(abaca), 짚(straw), 잎(leaves), 잔디 부스러기(grass clippings), 옥수수대(corn stover), 옥수수 속대(corn cobs), 증류립(distillers grains), 콩과 식물(legume plants), 수수(sorghum), 사탕수수(sugar cane), 사탕무 펄프(sugar beet pulp), 우드칩(wood chips), 톱밥(sawdust) 및 바이오매스 크롭(biomass crops)으로 이루어진 군으로부터 선택된 방법.
Applications Claiming Priority (5)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US27183309P | 2009-07-24 | 2009-07-24 | |
| US61/271,833 | 2009-07-24 | ||
| US28552609P | 2009-12-10 | 2009-12-10 | |
| US61/285,526 | 2009-12-10 | ||
| PCT/US2010/043279 WO2011011796A2 (en) | 2009-07-24 | 2010-07-26 | Methods and compositions for improving sugar transport, mixed sugar fermentation, and production of biofuels |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20120089631A true KR20120089631A (ko) | 2012-08-13 |
Family
ID=43497647
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020127002889A KR20120089631A (ko) | 2009-07-24 | 2010-07-26 | 개선된 당 수송, 혼합된 당 발효 및 바이오 연료의 생산을 위한 방법 및 조성물 |
Country Status (13)
| Country | Link |
|---|---|
| US (4) | US8431360B2 (ko) |
| EP (1) | EP2456878A4 (ko) |
| JP (1) | JP2013500019A (ko) |
| KR (1) | KR20120089631A (ko) |
| CN (1) | CN102625844A (ko) |
| AU (1) | AU2010275388A1 (ko) |
| BR (1) | BR112012001664A2 (ko) |
| CA (1) | CA2765313A1 (ko) |
| EA (1) | EA201290068A1 (ko) |
| IN (1) | IN2012DN00345A (ko) |
| MX (1) | MX2012001071A (ko) |
| NZ (1) | NZ597001A (ko) |
| WO (1) | WO2011011796A2 (ko) |
Families Citing this family (40)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| AU2010275388A1 (en) * | 2009-07-24 | 2012-01-12 | Bp Corporation North America Inc. | Methods and compositions for improving sugar transport, mixed sugar fermentation, and production of biofuels |
| US20120270290A1 (en) * | 2009-09-03 | 2012-10-25 | Kyoto University | Pentose transporter |
| WO2011123715A1 (en) * | 2010-03-31 | 2011-10-06 | The United States Of America As Represented By The Secretary Of Agriculture | Metabolically engineered yeasts for the production of ethanol and other products from xylose and cellobiose |
| WO2011161685A2 (en) | 2010-06-26 | 2011-12-29 | Hcl Cleantech Ltd. | Sugar mixtures and methods for production and use thereof |
| IL206678A0 (en) | 2010-06-28 | 2010-12-30 | Hcl Cleantech Ltd | A method for the production of fermentable sugars |
| IL207945A0 (en) | 2010-09-02 | 2010-12-30 | Robert Jansen | Method for the production of carbohydrates |
| US9096859B2 (en) | 2011-01-26 | 2015-08-04 | The Regents Of The University Of California | Microbial conversion of plant biomass to advanced biofuels |
| CN103597084A (zh) | 2011-02-07 | 2014-02-19 | 加利福尼亚大学董事会 | 增强的纤维糊精代谢 |
| GB2524906B8 (en) | 2011-04-07 | 2016-12-07 | Virdia Ltd | Lignocellulose conversion processes and products |
| CA2832587A1 (en) | 2011-04-11 | 2012-10-18 | Cargill, Incorporated | Compositions and methods for increased ethanol production from biomass |
| WO2012143513A2 (en) * | 2011-04-22 | 2012-10-26 | Dsm Ip Assets B.V. | Yeast cell capable of converting sugars including arabinose and xylose |
| US9617608B2 (en) | 2011-10-10 | 2017-04-11 | Virdia, Inc. | Sugar compositions |
| WO2013059326A1 (en) * | 2011-10-17 | 2013-04-25 | The Board Of Trustees Of The University Of Illinois | Xylitol production from cellulosic biomass |
| WO2013155481A1 (en) * | 2012-04-13 | 2013-10-17 | The Board Of Trustees Of The University Of Illinois | Improved cellodextrin transport and mixed sugar fermentation |
| SG11201407183SA (en) | 2012-05-03 | 2014-12-30 | Virdia Ltd | Methods for treating lignocellulosic materials |
| CN103060390A (zh) * | 2012-08-29 | 2013-04-24 | 太仓市茂通化建有限公司 | 一种利用玉米秸秆水解液燃料丁醇的方法 |
| CN104140977B (zh) * | 2013-05-07 | 2018-10-23 | 中国科学院天津工业生物技术研究所 | Cdt-2的新用途以及利用其促进微生物细胞转运木寡糖的方法及其应用 |
| US9926347B2 (en) | 2013-11-05 | 2018-03-27 | Board Of Regents, The University Of Texas System | Methods for engineering sugar transporter preferences |
| CN104844698B (zh) * | 2014-02-16 | 2020-09-25 | 中国科学院天津工业生物技术研究所 | 一种促进微生物细胞转运葡萄糖、木糖和阿拉伯糖的方法及其在生物基产品发酵中的应用 |
| JP6092157B2 (ja) * | 2014-06-09 | 2017-03-08 | 富士フイルム株式会社 | 電子機器、電子機器の表示制御方法、及び電子機器の表示制御プログラム |
| WO2015200281A1 (en) * | 2014-06-24 | 2015-12-30 | The Regents Of The University Of California | Methods for production of xylosyl-xylitol oligomers |
| WO2016012429A1 (en) * | 2014-07-24 | 2016-01-28 | Dsm Ip Assets B.V. | Yeast cell with improved pentose transport |
| JP6500406B2 (ja) * | 2014-12-01 | 2019-04-17 | セイコーエプソン株式会社 | 入出力制御装置、入出力制御プログラム |
| US11078548B2 (en) | 2015-01-07 | 2021-08-03 | Virdia, Llc | Method for producing xylitol by fermentation |
| WO2016145331A1 (en) * | 2015-03-12 | 2016-09-15 | Thermo Finnigan Llc | Methods for data-dependent mass spectrometry of mixed biomolecular analytes |
| BR112017025322A8 (pt) | 2015-05-27 | 2022-08-23 | Virdia Inc | Processos integrados para recuperação de hidrolisato celulósico após hidrólise de polpa de celulose |
| CN107236757A (zh) * | 2016-03-28 | 2017-10-10 | 中国科学院天津工业生物技术研究所 | 一种提高丝状真菌木质纤维素酶系表达及生物基化学品生产的方法 |
| WO2018027181A1 (en) | 2016-08-05 | 2018-02-08 | Kuehnle Agrosystems, Inc. | Producing and altering microbial fermentation products using non-commonly used lignocellulosic hydrolysates |
| BR112019004929A2 (pt) | 2016-09-13 | 2019-06-04 | Allergan Inc | composições de toxina clostridial não proteíca |
| JP2020526229A (ja) | 2017-06-30 | 2020-08-31 | ジミテック,インコーポレーテッド | オリゴ糖の使用向上のために操作された微生物 |
| AU2018327090A1 (en) * | 2017-08-30 | 2020-02-20 | Riken | Novel beta-glucosidase, enzyme composition including same, and method for manufacturing sugar solution using same |
| EP3746545A1 (en) * | 2018-01-29 | 2020-12-09 | Novozymes A/S | Microorganisms with improved nitrogen utilization for ethanol production |
| EP3543343B1 (en) * | 2018-03-12 | 2020-04-29 | INDIAN OIL CORPORATION Ltd. | Two-stage simultaneous saccharification and co-fermentation for producing ethanol from lignocellulose |
| CN110240271B (zh) * | 2018-04-28 | 2022-06-17 | 华东理工大学 | 一种处理含磷酸三乙酯废水的生物反应器的启动方法 |
| CN110982850B (zh) * | 2018-12-25 | 2022-07-29 | 华侨大学 | 一种半纤维素糖化能力增强的米曲霉工程菌合成木糖醇的方法 |
| CN109837232A (zh) * | 2019-03-27 | 2019-06-04 | 黑龙江大学 | 一种粗糙脉胞菌外切葡聚糖酶突变菌株的构建方法和应用 |
| CN111593014B (zh) * | 2020-06-24 | 2022-07-05 | 江南大学 | 联产1,3-丙二醇和d-1,2,4-丁三醇的方法 |
| CN112322563A (zh) * | 2020-10-20 | 2021-02-05 | 广西科学院 | 一种纤维素发酵工程菌及其构建方法和应用 |
| WO2022272119A2 (en) * | 2021-06-25 | 2022-12-29 | The Board Of Trustees Of The University Of Illinois | Synthetic toolkit for plant transformation |
| CN117821489A (zh) * | 2023-12-14 | 2024-04-05 | 湖北大学 | 一种利用重组酵母全细胞降解可再生塑料的方法 |
Family Cites Families (7)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US5100791A (en) | 1991-01-16 | 1992-03-31 | The United States Of America As Represented By The United States Department Of Energy | Simultaneous saccharification and fermentation (SSF) using cellobiose fermenting yeast Brettanomyces custersii |
| CN1261913A (zh) | 1997-07-04 | 2000-08-02 | 诺沃挪第克公司 | 来自糖丝菌的内切-β-1,4-葡聚糖酶 |
| US7344876B2 (en) | 2003-01-24 | 2008-03-18 | Phage Biotechnology, Inc. | Kluyveromyces strains metabolizing cellulosic and hemicellulosic materials |
| PT103331A (pt) * | 2005-08-05 | 2007-02-28 | Fundacao Da Faculdade De Cienc | Expressão dum transportador activo de xilose em saccharomyces cerevisiae modificada geneticamente |
| CN101310017B (zh) * | 2005-11-16 | 2013-03-20 | 诺维信公司 | 具有内切葡聚糖酶活性的多肽和编码该多肽的多核苷酸 |
| WO2008155665A2 (en) | 2007-05-09 | 2008-12-24 | University Of Stellenbosch | Method for enhancing cellobiose utilization |
| AU2010275388A1 (en) * | 2009-07-24 | 2012-01-12 | Bp Corporation North America Inc. | Methods and compositions for improving sugar transport, mixed sugar fermentation, and production of biofuels |
-
2010
- 2010-07-26 AU AU2010275388A patent/AU2010275388A1/en not_active Abandoned
- 2010-07-26 EA EA201290068A patent/EA201290068A1/ru unknown
- 2010-07-26 CN CN2010800398589A patent/CN102625844A/zh active Pending
- 2010-07-26 CA CA2765313A patent/CA2765313A1/en not_active Abandoned
- 2010-07-26 MX MX2012001071A patent/MX2012001071A/es active IP Right Grant
- 2010-07-26 JP JP2012521881A patent/JP2013500019A/ja active Pending
- 2010-07-26 NZ NZ597001A patent/NZ597001A/en not_active IP Right Cessation
- 2010-07-26 EP EP10803022A patent/EP2456878A4/en not_active Withdrawn
- 2010-07-26 KR KR1020127002889A patent/KR20120089631A/ko not_active Application Discontinuation
- 2010-07-26 IN IN345DEN2012 patent/IN2012DN00345A/en unknown
- 2010-07-26 WO PCT/US2010/043279 patent/WO2011011796A2/en active Application Filing
- 2010-07-26 BR BR112012001664A patent/BR112012001664A2/pt not_active IP Right Cessation
- 2010-07-26 US US12/843,844 patent/US8431360B2/en not_active Expired - Fee Related
-
2013
- 2013-03-13 US US13/802,533 patent/US8765410B2/en not_active Expired - Fee Related
-
2014
- 2014-05-28 US US14/289,570 patent/US9012177B2/en not_active Expired - Fee Related
-
2015
- 2015-01-28 US US14/608,018 patent/US20150299755A1/en not_active Abandoned
Also Published As
| Publication number | Publication date |
|---|---|
| US20130316437A1 (en) | 2013-11-28 |
| EA201290068A1 (ru) | 2013-05-30 |
| EP2456878A2 (en) | 2012-05-30 |
| US8765410B2 (en) | 2014-07-01 |
| BR112012001664A2 (pt) | 2018-12-26 |
| US9012177B2 (en) | 2015-04-21 |
| MX2012001071A (es) | 2012-10-09 |
| WO2011011796A3 (en) | 2011-09-09 |
| AU2010275388A8 (en) | 2012-06-14 |
| US20110020910A1 (en) | 2011-01-27 |
| IN2012DN00345A (ko) | 2015-05-08 |
| AU2010275388A1 (en) | 2012-01-12 |
| CN102625844A (zh) | 2012-08-01 |
| CA2765313A1 (en) | 2011-01-27 |
| WO2011011796A8 (en) | 2011-12-22 |
| US20150010985A1 (en) | 2015-01-08 |
| WO2011011796A2 (en) | 2011-01-27 |
| US8431360B2 (en) | 2013-04-30 |
| JP2013500019A (ja) | 2013-01-07 |
| EP2456878A4 (en) | 2013-01-02 |
| US20150299755A1 (en) | 2015-10-22 |
| WO2011011796A9 (en) | 2011-06-23 |
| NZ597001A (en) | 2014-05-30 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| US9012177B2 (en) | Methods and compositions for improving sugar transport, mixed sugar fermentation, and production of biofuels | |
| DK2748317T3 (en) | GH61 glycoside hydrolase protein variants and cofactors that enhance GH61 activity | |
| CA3021166C (en) | Heterologous expression of fungal cellobiohydrolases in yeast | |
| DK2361311T3 (en) | MAKE EXPRESSIVE CELLULASES FOR SIMULTANEOUS INSURANCE AND ACTION BY CELLULOSE | |
| EP3168300A1 (en) | Yeast expressing saccharolytic enzymes for consolidated bioprocessing using starch and cellulose | |
| US20130040297A1 (en) | Process for the production of cells which are capable of converting arabinose | |
| CN117925431A (zh) | 改进的无甘油的乙醇生产 | |
| AU2012214520A1 (en) | Enhanced cellodextrin metabolism | |
| CA3077115A1 (en) | Improved glycerol free ethanol production | |
| WO2013071112A1 (en) | A genetically modified strain of s. cerevisiae engineered to ferment xylose and arabinose | |
| US20140342418A1 (en) | Xylitol production from cellulosic biomass | |
| EP3332004A1 (en) | Xylose isomerase-modified yeast strains and methods for bioproduct production | |
| WO2013155481A1 (en) | Improved cellodextrin transport and mixed sugar fermentation | |
| US11091753B2 (en) | Xylose fermenting yeast strains and processes thereof for ethanol production | |
| US20140154751A1 (en) | Enhanced fermentation of cellodextrins and beta-d-glucose | |
| US9670515B2 (en) | Methods for production of xylosylxylitol oligomers | |
| BR112021012497A2 (pt) | Levedura recombinante e método para produzir etanol usando a mesma | |
| Class et al. | Patent application title: METHODS AND COMPOSITIONS FOR IMPROVING SUGAR TRANSPORT, MIXED SUGAR FERMENTATION, AND PRODUCTION OF BIOFUELS | |
| CN114507608B (zh) | 一种在线粒体内生产乙醇的重组丝状真菌及其构建和应用 | |
| WO2023079048A1 (en) | Process for the production of ethanol and recombinant yeast cell |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| WITN | Application deemed withdrawn, e.g. because no request for examination was filed or no examination fee was paid |