JP2013500019A - 糖輸送、混合糖の発酵および生物燃料の産生を促進させる方法ならびに組成物 - Google Patents
糖輸送、混合糖の発酵および生物燃料の産生を促進させる方法ならびに組成物 Download PDFInfo
- Publication number
- JP2013500019A JP2013500019A JP2012521881A JP2012521881A JP2013500019A JP 2013500019 A JP2013500019 A JP 2013500019A JP 2012521881 A JP2012521881 A JP 2012521881A JP 2012521881 A JP2012521881 A JP 2012521881A JP 2013500019 A JP2013500019 A JP 2013500019A
- Authority
- JP
- Japan
- Prior art keywords
- helix
- transmembrane
- host cell
- recombinant polynucleotide
- polypeptide
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Pending
Links
- 238000000034 method Methods 0.000 title claims abstract description 315
- 230000001737 promoting effect Effects 0.000 title claims abstract description 101
- 235000000346 sugar Nutrition 0.000 title claims abstract description 58
- 238000000855 fermentation Methods 0.000 title claims description 40
- 239000000203 mixture Substances 0.000 title claims description 31
- 238000004519 manufacturing process Methods 0.000 title abstract description 22
- 230000004151 fermentation Effects 0.000 title description 31
- 239000002551 biofuel Substances 0.000 title description 5
- 102000040430 polynucleotide Human genes 0.000 claims abstract description 513
- 108091033319 polynucleotide Proteins 0.000 claims abstract description 513
- 239000002157 polynucleotide Substances 0.000 claims abstract description 513
- 108090000765 processed proteins & peptides Proteins 0.000 claims abstract description 281
- 102000004196 processed proteins & peptides Human genes 0.000 claims abstract description 278
- 229920001184 polypeptide Polymers 0.000 claims abstract description 277
- 229930195733 hydrocarbon Natural products 0.000 claims abstract description 213
- 150000002430 hydrocarbons Chemical class 0.000 claims abstract description 213
- 239000004215 Carbon black (E152) Substances 0.000 claims abstract description 173
- 230000032258 transport Effects 0.000 claims abstract description 173
- FYGDTMLNYKFZSV-ZWSAEMDYSA-N cellotriose Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)O[C@H]1O[C@@H]1[C@@H](CO)O[C@@H](O[C@@H]2[C@H](OC(O)[C@H](O)[C@H]2O)CO)[C@H](O)[C@H]1O FYGDTMLNYKFZSV-ZWSAEMDYSA-N 0.000 claims abstract description 154
- 229920002299 Cellodextrin Polymers 0.000 claims abstract description 139
- 150000002972 pentoses Chemical class 0.000 claims abstract description 61
- 230000010261 cell growth Effects 0.000 claims abstract description 46
- 229920002678 cellulose Polymers 0.000 claims abstract description 38
- 239000001913 cellulose Substances 0.000 claims abstract description 38
- 229920002488 Hemicellulose Polymers 0.000 claims abstract description 19
- SRBFZHDQGSBBOR-IOVATXLUSA-N Xylose Natural products O[C@@H]1COC(O)[C@H](O)[C@H]1O SRBFZHDQGSBBOR-IOVATXLUSA-N 0.000 claims description 291
- SRBFZHDQGSBBOR-UHFFFAOYSA-N beta-D-Pyranose-Lyxose Natural products OC1COC(O)C(O)C1O SRBFZHDQGSBBOR-UHFFFAOYSA-N 0.000 claims description 217
- PYMYPHUHKUWMLA-UHFFFAOYSA-N arabinose Natural products OCC(O)C(O)C(O)C=O PYMYPHUHKUWMLA-UHFFFAOYSA-N 0.000 claims description 212
- 108090000623 proteins and genes Proteins 0.000 claims description 135
- 230000014509 gene expression Effects 0.000 claims description 121
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 claims description 100
- WQZGKKKJIJFFOK-VFUOTHLCSA-N beta-D-glucose Chemical compound OC[C@H]1O[C@@H](O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-VFUOTHLCSA-N 0.000 claims description 99
- 239000008103 glucose Substances 0.000 claims description 99
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 claims description 97
- 238000012258 culturing Methods 0.000 claims description 92
- 238000003786 synthesis reaction Methods 0.000 claims description 81
- 230000015572 biosynthetic process Effects 0.000 claims description 80
- 108010078791 Carrier Proteins Proteins 0.000 claims description 73
- GUBGYTABKSRVRQ-CUHNMECISA-N D-Cellobiose Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)O[C@H]1O[C@@H]1[C@@H](CO)OC(O)[C@H](O)[C@H]1O GUBGYTABKSRVRQ-CUHNMECISA-N 0.000 claims description 69
- PYMYPHUHKUWMLA-WDCZJNDASA-N arabinose Chemical compound OC[C@@H](O)[C@@H](O)[C@H](O)C=O PYMYPHUHKUWMLA-WDCZJNDASA-N 0.000 claims description 66
- 102000004190 Enzymes Human genes 0.000 claims description 63
- 108090000790 Enzymes Proteins 0.000 claims description 63
- 229940088598 enzyme Drugs 0.000 claims description 63
- 108010047754 beta-Glucosidase Proteins 0.000 claims description 62
- 102000006995 beta-Glucosidase Human genes 0.000 claims description 61
- 241000221961 Neurospora crassa Species 0.000 claims description 59
- 230000000694 effects Effects 0.000 claims description 51
- 240000004808 Saccharomyces cerevisiae Species 0.000 claims description 48
- 235000014680 Saccharomyces cerevisiae Nutrition 0.000 claims description 48
- 241001123227 Saccharomyces pastorianus Species 0.000 claims description 34
- 150000001413 amino acids Chemical class 0.000 claims description 34
- 241000235060 Scheffersomyces stipitis Species 0.000 claims description 31
- 108010059892 Cellulase Proteins 0.000 claims description 30
- 229940106157 cellulase Drugs 0.000 claims description 27
- 230000003197 catalytic effect Effects 0.000 claims description 26
- 239000002028 Biomass Substances 0.000 claims description 22
- 229920000642 polymer Polymers 0.000 claims description 22
- 101100393881 Arabidopsis thaliana GT16 gene Proteins 0.000 claims description 21
- 102000016912 Aldehyde Reductase Human genes 0.000 claims description 18
- 108010053754 Aldehyde reductase Proteins 0.000 claims description 18
- 241000235070 Saccharomyces Species 0.000 claims description 17
- LUEWUZLMQUOBSB-ZLBHSGTGSA-N alpha-maltotetraose Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)O[C@@H]1O[C@@H]1[C@@H](CO)O[C@H](O[C@@H]2[C@H](O[C@H](O[C@@H]3[C@H](O[C@H](O)[C@H](O)[C@H]3O)CO)[C@H](O)[C@H]2O)CO)[C@H](O)[C@H]1O LUEWUZLMQUOBSB-ZLBHSGTGSA-N 0.000 claims description 17
- 241000193464 Clostridium sp. Species 0.000 claims description 16
- 241000193448 Ruminiclostridium thermocellum Species 0.000 claims description 16
- 241000192263 Scheffersomyces shehatae Species 0.000 claims description 16
- 241001313536 Thermothelomyces thermophila Species 0.000 claims description 16
- 241000588902 Zymomonas mobilis Species 0.000 claims description 16
- 102100035263 Anion exchange transporter Human genes 0.000 claims description 15
- 101001094042 Homo sapiens Anion exchange transporter Proteins 0.000 claims description 15
- 241000933069 Lachnoclostridium phytofermentans Species 0.000 claims description 15
- 101150053912 SUT3 gene Proteins 0.000 claims description 15
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 claims description 14
- 108010058076 D-xylulose reductase Proteins 0.000 claims description 14
- 241000588724 Escherichia coli Species 0.000 claims description 14
- 241000233866 Fungi Species 0.000 claims description 14
- 108010064700 L-arabinitol 4-dehydrogenase Proteins 0.000 claims description 14
- 102100026974 Sorbitol dehydrogenase Human genes 0.000 claims description 14
- 241000196324 Embryophyta Species 0.000 claims description 13
- 102100030931 Ladinin-1 Human genes 0.000 claims description 13
- 108700040099 Xylose isomerases Proteins 0.000 claims description 13
- 102100029089 Xylulose kinase Human genes 0.000 claims description 13
- 108091022915 xylulokinase Proteins 0.000 claims description 13
- 241000193454 Clostridium beijerinckii Species 0.000 claims description 12
- 241000235072 Saccharomyces bayanus Species 0.000 claims description 12
- 241000235088 Saccharomyces sp. Species 0.000 claims description 12
- 241000193401 Clostridium acetobutylicum Species 0.000 claims description 11
- 108010018080 L-arabinose isomerase Proteins 0.000 claims description 11
- FNZLKVNUWIIPSJ-CRCLSJGQSA-L L-ribulose 5-phosphate(2-) Chemical compound OCC(=O)[C@@H](O)[C@@H](O)COP([O-])([O-])=O FNZLKVNUWIIPSJ-CRCLSJGQSA-L 0.000 claims description 11
- 108090001066 Racemases and epimerases Proteins 0.000 claims description 11
- 235000003534 Saccharomyces carlsbergensis Nutrition 0.000 claims description 11
- 230000002829 reductive effect Effects 0.000 claims description 11
- 241001138401 Kluyveromyces lactis Species 0.000 claims description 10
- 241000193459 Moorella thermoacetica Species 0.000 claims description 10
- 239000000446 fuel Substances 0.000 claims description 10
- 241000170280 Kluyveromyces sp. Species 0.000 claims description 9
- 108010002430 hemicellulase Proteins 0.000 claims description 9
- 241000222178 Candida tropicalis Species 0.000 claims description 8
- 241000235649 Kluyveromyces Species 0.000 claims description 8
- 244000285963 Kluyveromyces fragilis Species 0.000 claims description 8
- 235000014663 Kluyveromyces fragilis Nutrition 0.000 claims description 8
- 108010080643 L-xylulose reductase Proteins 0.000 claims description 8
- 102100029137 L-xylulose reductase Human genes 0.000 claims description 8
- LRHPLDYGYMQRHN-UHFFFAOYSA-N N-Butanol Chemical compound CCCCO LRHPLDYGYMQRHN-UHFFFAOYSA-N 0.000 claims description 8
- 230000001939 inductive effect Effects 0.000 claims description 8
- 108020002667 ribulokinase Proteins 0.000 claims description 8
- 241000894006 Bacteria Species 0.000 claims description 7
- 241000588749 Klebsiella oxytoca Species 0.000 claims description 7
- 229940059442 hemicellulase Drugs 0.000 claims description 7
- 239000000463 material Substances 0.000 claims description 7
- 241000222120 Candida <Saccharomycetales> Species 0.000 claims description 6
- 101100216047 Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) gla-1 gene Proteins 0.000 claims description 5
- 240000008042 Zea mays Species 0.000 claims description 5
- 235000005824 Zea mays ssp. parviglumis Nutrition 0.000 claims description 5
- 235000002017 Zea mays subsp mays Nutrition 0.000 claims description 5
- 235000005822 corn Nutrition 0.000 claims description 5
- 230000035772 mutation Effects 0.000 claims description 5
- 101710088194 Dehydrogenase Proteins 0.000 claims description 4
- 241001137870 Thermoanaerobacterium Species 0.000 claims description 3
- 244000198134 Agave sisalana Species 0.000 claims description 2
- 241000609240 Ambelania acida Species 0.000 claims description 2
- 235000017166 Bambusa arundinacea Nutrition 0.000 claims description 2
- 235000017491 Bambusa tulda Nutrition 0.000 claims description 2
- 241000219310 Beta vulgaris subsp. vulgaris Species 0.000 claims description 2
- 235000004431 Linum usitatissimum Nutrition 0.000 claims description 2
- 240000006240 Linum usitatissimum Species 0.000 claims description 2
- 240000000907 Musa textilis Species 0.000 claims description 2
- 240000007594 Oryza sativa Species 0.000 claims description 2
- 235000007164 Oryza sativa Nutrition 0.000 claims description 2
- 241001520808 Panicum virgatum Species 0.000 claims description 2
- 235000015334 Phyllostachys viridis Nutrition 0.000 claims description 2
- 240000000111 Saccharum officinarum Species 0.000 claims description 2
- 235000007201 Saccharum officinarum Nutrition 0.000 claims description 2
- 235000021536 Sugar beet Nutrition 0.000 claims description 2
- 239000010905 bagasse Substances 0.000 claims description 2
- 239000011425 bamboo Substances 0.000 claims description 2
- 235000013339 cereals Nutrition 0.000 claims description 2
- 239000000835 fiber Substances 0.000 claims description 2
- 238000003306 harvesting Methods 0.000 claims description 2
- 239000010903 husk Substances 0.000 claims description 2
- 235000021374 legumes Nutrition 0.000 claims description 2
- 235000009566 rice Nutrition 0.000 claims description 2
- 239000010902 straw Substances 0.000 claims description 2
- 125000000969 xylosyl group Chemical group C1([C@H](O)[C@@H](O)[C@H](O)CO1)* 0.000 claims description 2
- 235000020004 porter Nutrition 0.000 claims 5
- 108010056771 Glucosidases Proteins 0.000 claims 1
- 102000004366 Glucosidases Human genes 0.000 claims 1
- 240000003483 Leersia hexandra Species 0.000 claims 1
- 240000003433 Miscanthus floridulus Species 0.000 claims 1
- 244000082204 Phyllostachys viridis Species 0.000 claims 1
- 240000006394 Sorghum bicolor Species 0.000 claims 1
- 235000011684 Sorghum saccharatum Nutrition 0.000 claims 1
- 239000002773 nucleotide Substances 0.000 abstract description 16
- 125000003729 nucleotide group Chemical group 0.000 abstract description 16
- 230000002708 enhancing effect Effects 0.000 abstract 1
- 210000004027 cell Anatomy 0.000 description 405
- 239000002609 medium Substances 0.000 description 78
- 102000004169 proteins and genes Human genes 0.000 description 73
- 235000018102 proteins Nutrition 0.000 description 70
- 150000007523 nucleic acids Chemical class 0.000 description 41
- 239000013604 expression vector Substances 0.000 description 28
- 235000010980 cellulose Nutrition 0.000 description 26
- 102000039446 nucleic acids Human genes 0.000 description 26
- 108020004707 nucleic acids Proteins 0.000 description 26
- 235000001014 amino acid Nutrition 0.000 description 24
- 229940024606 amino acid Drugs 0.000 description 24
- 241000221960 Neurospora Species 0.000 description 20
- 239000012528 membrane Substances 0.000 description 19
- 230000008859 change Effects 0.000 description 17
- 150000008163 sugars Chemical class 0.000 description 17
- 230000012010 growth Effects 0.000 description 16
- 241000894007 species Species 0.000 description 15
- OKTJSMMVPCPJKN-UHFFFAOYSA-N Carbon Chemical compound [C] OKTJSMMVPCPJKN-UHFFFAOYSA-N 0.000 description 14
- 241000878007 Miscanthus Species 0.000 description 14
- 229910052799 carbon Inorganic materials 0.000 description 14
- 101100438645 Saccharomyces cerevisiae (strain ATCC 204508 / S288c) CBT1 gene Proteins 0.000 description 13
- 230000006870 function Effects 0.000 description 13
- 239000000178 monomer Substances 0.000 description 13
- 108091028043 Nucleic acid sequence Proteins 0.000 description 12
- 238000004458 analytical method Methods 0.000 description 12
- 230000009471 action Effects 0.000 description 11
- 125000000539 amino acid group Chemical group 0.000 description 11
- 238000012217 deletion Methods 0.000 description 11
- 230000037430 deletion Effects 0.000 description 11
- 238000002474 experimental method Methods 0.000 description 11
- 239000005090 green fluorescent protein Substances 0.000 description 11
- 230000004048 modification Effects 0.000 description 11
- 238000012986 modification Methods 0.000 description 11
- 230000037361 pathway Effects 0.000 description 11
- 239000013612 plasmid Substances 0.000 description 11
- 239000000706 filtrate Substances 0.000 description 10
- 230000002538 fungal effect Effects 0.000 description 10
- 150000001875 compounds Chemical class 0.000 description 9
- 230000007423 decrease Effects 0.000 description 9
- 239000001963 growth medium Substances 0.000 description 9
- 230000003834 intracellular effect Effects 0.000 description 9
- 244000005700 microbiome Species 0.000 description 9
- 108010034634 Repressor Proteins Proteins 0.000 description 8
- 102000009661 Repressor Proteins Human genes 0.000 description 8
- 230000001976 improved effect Effects 0.000 description 8
- 230000002018 overexpression Effects 0.000 description 8
- 230000035515 penetration Effects 0.000 description 8
- 238000006467 substitution reaction Methods 0.000 description 8
- 241000588722 Escherichia Species 0.000 description 7
- 230000004071 biological effect Effects 0.000 description 7
- 230000015556 catabolic process Effects 0.000 description 7
- 238000012239 gene modification Methods 0.000 description 7
- 230000005017 genetic modification Effects 0.000 description 7
- 235000013617 genetically modified food Nutrition 0.000 description 7
- 230000000149 penetrating effect Effects 0.000 description 7
- 230000000153 supplemental effect Effects 0.000 description 7
- 239000013598 vector Substances 0.000 description 7
- 108020004414 DNA Proteins 0.000 description 6
- ISWSIDIOOBJBQZ-UHFFFAOYSA-N Phenol Chemical compound OC1=CC=CC=C1 ISWSIDIOOBJBQZ-UHFFFAOYSA-N 0.000 description 6
- 102000004879 Racemases and epimerases Human genes 0.000 description 6
- 238000003556 assay Methods 0.000 description 6
- -1 hexose sugars Chemical class 0.000 description 6
- 230000001965 increasing effect Effects 0.000 description 6
- 238000012360 testing method Methods 0.000 description 6
- 238000013518 transcription Methods 0.000 description 6
- 230000035897 transcription Effects 0.000 description 6
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 5
- 102000005575 Cellulases Human genes 0.000 description 5
- 108010084185 Cellulases Proteins 0.000 description 5
- 108010043121 Green Fluorescent Proteins Proteins 0.000 description 5
- 102000004144 Green Fluorescent Proteins Human genes 0.000 description 5
- 241000235648 Pichia Species 0.000 description 5
- 238000007792 addition Methods 0.000 description 5
- 230000001186 cumulative effect Effects 0.000 description 5
- 238000003752 polymerase chain reaction Methods 0.000 description 5
- 230000001105 regulatory effect Effects 0.000 description 5
- 108091008146 restriction endonucleases Proteins 0.000 description 5
- OWEGMIWEEQEYGQ-UHFFFAOYSA-N 100676-05-9 Natural products OC1C(O)C(O)C(CO)OC1OCC1C(O)C(O)C(O)C(OC2C(OC(O)C(O)C2O)CO)O1 OWEGMIWEEQEYGQ-UHFFFAOYSA-N 0.000 description 4
- UHPMCKVQTMMPCG-UHFFFAOYSA-N 5,8-dihydroxy-2-methoxy-6-methyl-7-(2-oxopropyl)naphthalene-1,4-dione Chemical compound CC1=C(CC(C)=O)C(O)=C2C(=O)C(OC)=CC(=O)C2=C1O UHPMCKVQTMMPCG-UHFFFAOYSA-N 0.000 description 4
- 101100272861 Arabidopsis thaliana BXL3 gene Proteins 0.000 description 4
- 241000223218 Fusarium Species 0.000 description 4
- DHMQDGOQFOQNFH-UHFFFAOYSA-N Glycine Chemical compound NCC(O)=O DHMQDGOQFOQNFH-UHFFFAOYSA-N 0.000 description 4
- GUBGYTABKSRVRQ-PICCSMPSSA-N Maltose Natural products O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)O[C@@H]1O[C@@H]1[C@@H](CO)OC(O)[C@H](O)[C@H]1O GUBGYTABKSRVRQ-PICCSMPSSA-N 0.000 description 4
- 241000178985 Moorella Species 0.000 description 4
- 102000004316 Oxidoreductases Human genes 0.000 description 4
- 108090000854 Oxidoreductases Proteins 0.000 description 4
- 108091023040 Transcription factor Proteins 0.000 description 4
- 102000040945 Transcription factor Human genes 0.000 description 4
- 101150032931 XYL3 gene Proteins 0.000 description 4
- GUBGYTABKSRVRQ-QUYVBRFLSA-N beta-maltose Chemical compound OC[C@H]1O[C@H](O[C@H]2[C@H](O)[C@@H](O)[C@H](O)O[C@@H]2CO)[C@H](O)[C@@H](O)[C@@H]1O GUBGYTABKSRVRQ-QUYVBRFLSA-N 0.000 description 4
- 210000002421 cell wall Anatomy 0.000 description 4
- 230000003247 decreasing effect Effects 0.000 description 4
- 238000006731 degradation reaction Methods 0.000 description 4
- 230000001419 dependent effect Effects 0.000 description 4
- 239000012634 fragment Substances 0.000 description 4
- 239000000499 gel Substances 0.000 description 4
- 230000002401 inhibitory effect Effects 0.000 description 4
- 230000004807 localization Effects 0.000 description 4
- 238000005259 measurement Methods 0.000 description 4
- 210000004940 nucleus Anatomy 0.000 description 4
- 239000000047 product Substances 0.000 description 4
- 239000007787 solid Substances 0.000 description 4
- QTBSBXVTEAMEQO-UHFFFAOYSA-N Acetic acid Chemical compound CC(O)=O QTBSBXVTEAMEQO-UHFFFAOYSA-N 0.000 description 3
- GUBGYTABKSRVRQ-XLOQQCSPSA-N Alpha-Lactose Chemical compound O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@H]1O[C@@H]1[C@@H](CO)O[C@H](O)[C@H](O)[C@H]1O GUBGYTABKSRVRQ-XLOQQCSPSA-N 0.000 description 3
- 241000221779 Fusarium sambucinum Species 0.000 description 3
- WHUUTDBJXJRKMK-UHFFFAOYSA-N Glutamic acid Natural products OC(=O)C(N)CCC(O)=O WHUUTDBJXJRKMK-UHFFFAOYSA-N 0.000 description 3
- CKLJMWTZIZZHCS-REOHCLBHSA-N L-aspartic acid Chemical compound OC(=O)[C@@H](N)CC(O)=O CKLJMWTZIZZHCS-REOHCLBHSA-N 0.000 description 3
- QIVBCDIJIAJPQS-VIFPVBQESA-N L-tryptophane Chemical compound C1=CC=C2C(C[C@H](N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-VIFPVBQESA-N 0.000 description 3
- GUBGYTABKSRVRQ-QKKXKWKRSA-N Lactose Natural products OC[C@H]1O[C@@H](O[C@H]2[C@H](O)[C@@H](O)C(O)O[C@@H]2CO)[C@H](O)[C@@H](O)[C@H]1O GUBGYTABKSRVRQ-QKKXKWKRSA-N 0.000 description 3
- 101000822885 Naja kaouthia Cobrotoxin II Proteins 0.000 description 3
- 108091005461 Nucleic proteins Proteins 0.000 description 3
- 101100069420 Saccharomyces cerevisiae (strain ATCC 204508 / S288c) GRE3 gene Proteins 0.000 description 3
- CZMRCDWAGMRECN-UGDNZRGBSA-N Sucrose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 CZMRCDWAGMRECN-UGDNZRGBSA-N 0.000 description 3
- 229930006000 Sucrose Natural products 0.000 description 3
- 241001137871 Thermoanaerobacterium saccharolyticum Species 0.000 description 3
- QIVBCDIJIAJPQS-UHFFFAOYSA-N Tryptophan Natural products C1=CC=C2C(CC(N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-UHFFFAOYSA-N 0.000 description 3
- OCIBBXPLUVYKCH-FYTDUCIRSA-N beta-D-cellohexaose Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)O[C@H]1O[C@@H]1[C@@H](CO)O[C@@H](O[C@@H]2[C@H](O[C@@H](O[C@@H]3[C@H](O[C@@H](O[C@@H]4[C@H](O[C@@H](O[C@@H]5[C@H](O[C@@H](O)[C@H](O)[C@H]5O)CO)[C@H](O)[C@H]4O)CO)[C@H](O)[C@H]3O)CO)[C@H](O)[C@H]2O)CO)[C@H](O)[C@H]1O OCIBBXPLUVYKCH-FYTDUCIRSA-N 0.000 description 3
- 238000004364 calculation method Methods 0.000 description 3
- 101150052795 cbh-1 gene Proteins 0.000 description 3
- 125000001547 cellobiose group Chemical group 0.000 description 3
- OCIBBXPLUVYKCH-UHFFFAOYSA-N cellopentanose Natural products OC1C(O)C(O)C(CO)OC1OC1C(CO)OC(OC2C(OC(OC3C(OC(OC4C(OC(OC5C(OC(O)C(O)C5O)CO)C(O)C4O)CO)C(O)C3O)CO)C(O)C2O)CO)C(O)C1O OCIBBXPLUVYKCH-UHFFFAOYSA-N 0.000 description 3
- 238000006243 chemical reaction Methods 0.000 description 3
- 238000010367 cloning Methods 0.000 description 3
- 230000000295 complement effect Effects 0.000 description 3
- 210000003527 eukaryotic cell Anatomy 0.000 description 3
- 230000002068 genetic effect Effects 0.000 description 3
- 230000007062 hydrolysis Effects 0.000 description 3
- 238000006460 hydrolysis reaction Methods 0.000 description 3
- 238000001727 in vivo Methods 0.000 description 3
- 238000011534 incubation Methods 0.000 description 3
- 239000008101 lactose Substances 0.000 description 3
- 239000002029 lignocellulosic biomass Substances 0.000 description 3
- 150000002772 monosaccharides Chemical class 0.000 description 3
- 239000000843 powder Substances 0.000 description 3
- 238000002415 sodium dodecyl sulfate polyacrylamide gel electrophoresis Methods 0.000 description 3
- 239000000758 substrate Substances 0.000 description 3
- 239000005720 sucrose Substances 0.000 description 3
- 230000001629 suppression Effects 0.000 description 3
- 230000014616 translation Effects 0.000 description 3
- 235000013311 vegetables Nutrition 0.000 description 3
- 101710129685 Arabinose-proton symporter Proteins 0.000 description 2
- 241000228212 Aspergillus Species 0.000 description 2
- 240000006439 Aspergillus oryzae Species 0.000 description 2
- 244000063299 Bacillus subtilis Species 0.000 description 2
- 235000014469 Bacillus subtilis Nutrition 0.000 description 2
- 241000221198 Basidiomycota Species 0.000 description 2
- 229920002134 Carboxymethyl cellulose Polymers 0.000 description 2
- 101710098246 Exoglucanase 2 Proteins 0.000 description 2
- 241000567163 Fusarium cerealis Species 0.000 description 2
- 241000146406 Fusarium heterosporum Species 0.000 description 2
- 239000004471 Glycine Substances 0.000 description 2
- 102000004157 Hydrolases Human genes 0.000 description 2
- 108090000604 Hydrolases Proteins 0.000 description 2
- FFEARJCKVFRZRR-BYPYZUCNSA-N L-methionine Chemical compound CSCC[C@H](N)C(O)=O FFEARJCKVFRZRR-BYPYZUCNSA-N 0.000 description 2
- 102000015841 Major facilitator superfamily Human genes 0.000 description 2
- 108050004064 Major facilitator superfamily Proteins 0.000 description 2
- DFPAKSUCGFBDDF-UHFFFAOYSA-N Nicotinamide Chemical compound NC(=O)C1=CC=CN=C1 DFPAKSUCGFBDDF-UHFFFAOYSA-N 0.000 description 2
- 241000233654 Oomycetes Species 0.000 description 2
- 241000228143 Penicillium Species 0.000 description 2
- 108020004511 Recombinant DNA Proteins 0.000 description 2
- 241000220317 Rosa Species 0.000 description 2
- 101100451954 Saccharomyces cerevisiae (strain ATCC 204508 / S288c) HXT1 gene Proteins 0.000 description 2
- 101100382629 Schizosaccharomyces pombe (strain 972 / ATCC 24843) cbh1 gene Proteins 0.000 description 2
- 241000499912 Trichoderma reesei Species 0.000 description 2
- 241000355691 Trichoderma reesei QM9414 Species 0.000 description 2
- 239000007983 Tris buffer Substances 0.000 description 2
- 241001148070 Vibrio furnissii Species 0.000 description 2
- 230000035508 accumulation Effects 0.000 description 2
- 238000009825 accumulation Methods 0.000 description 2
- DZBUGLKDJFMEHC-UHFFFAOYSA-N acridine Chemical compound C1=CC=CC2=CC3=CC=CC=C3N=C21 DZBUGLKDJFMEHC-UHFFFAOYSA-N 0.000 description 2
- 230000004913 activation Effects 0.000 description 2
- GZCGUPFRVQAUEE-SLPGGIOYSA-N aldehydo-D-glucose Chemical compound OC[C@@H](O)[C@@H](O)[C@H](O)[C@@H](O)C=O GZCGUPFRVQAUEE-SLPGGIOYSA-N 0.000 description 2
- PYMYPHUHKUWMLA-LMVFSUKVSA-N aldehydo-D-ribose Chemical compound OC[C@@H](O)[C@@H](O)[C@@H](O)C=O PYMYPHUHKUWMLA-LMVFSUKVSA-N 0.000 description 2
- PYMYPHUHKUWMLA-WISUUJSJSA-N aldehydo-L-xylose Chemical group OC[C@H](O)[C@@H](O)[C@H](O)C=O PYMYPHUHKUWMLA-WISUUJSJSA-N 0.000 description 2
- WQZGKKKJIJFFOK-PHYPRBDBSA-N alpha-D-galactose Chemical compound OC[C@H]1O[C@H](O)[C@H](O)[C@@H](O)[C@H]1O WQZGKKKJIJFFOK-PHYPRBDBSA-N 0.000 description 2
- 235000003704 aspartic acid Nutrition 0.000 description 2
- 230000001580 bacterial effect Effects 0.000 description 2
- 125000000188 beta-D-glucosyl group Chemical group C1([C@H](O)[C@@H](O)[C@H](O)[C@H](O1)CO)* 0.000 description 2
- OQFSQFPPLPISGP-UHFFFAOYSA-N beta-carboxyaspartic acid Natural products OC(=O)C(N)C(C(O)=O)C(O)=O OQFSQFPPLPISGP-UHFFFAOYSA-N 0.000 description 2
- 230000033228 biological regulation Effects 0.000 description 2
- 239000000872 buffer Substances 0.000 description 2
- 210000004899 c-terminal region Anatomy 0.000 description 2
- 238000004422 calculation algorithm Methods 0.000 description 2
- 101150048033 cbh gene Proteins 0.000 description 2
- FTNIPWXXIGNQQF-XHCCAYEESA-N cellopentaose Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)O[C@H]1O[C@@H]1[C@@H](CO)O[C@@H](O[C@@H]2[C@H](O[C@@H](O[C@@H]3[C@H](O[C@@H](O[C@@H]4[C@H](OC(O)[C@H](O)[C@H]4O)CO)[C@H](O)[C@H]3O)CO)[C@H](O)[C@H]2O)CO)[C@H](O)[C@H]1O FTNIPWXXIGNQQF-XHCCAYEESA-N 0.000 description 2
- 238000012512 characterization method Methods 0.000 description 2
- 238000010276 construction Methods 0.000 description 2
- 230000000875 corresponding effect Effects 0.000 description 2
- 238000011161 development Methods 0.000 description 2
- 230000018109 developmental process Effects 0.000 description 2
- 230000029087 digestion Effects 0.000 description 2
- 230000002255 enzymatic effect Effects 0.000 description 2
- 229930182830 galactose Natural products 0.000 description 2
- 230000004190 glucose uptake Effects 0.000 description 2
- 235000013922 glutamic acid Nutrition 0.000 description 2
- 239000004220 glutamic acid Substances 0.000 description 2
- 125000003147 glycosyl group Chemical group 0.000 description 2
- 125000002887 hydroxy group Chemical group [H]O* 0.000 description 2
- 230000006872 improvement Effects 0.000 description 2
- 238000010348 incorporation Methods 0.000 description 2
- 229910052738 indium Inorganic materials 0.000 description 2
- 230000006698 induction Effects 0.000 description 2
- 230000005764 inhibitory process Effects 0.000 description 2
- 230000000977 initiatory effect Effects 0.000 description 2
- JVTAAEKCZFNVCJ-UHFFFAOYSA-N lactic acid Chemical compound CC(O)C(O)=O JVTAAEKCZFNVCJ-UHFFFAOYSA-N 0.000 description 2
- 108010060845 lactose permease Proteins 0.000 description 2
- 239000011159 matrix material Substances 0.000 description 2
- 230000037353 metabolic pathway Effects 0.000 description 2
- 239000002207 metabolite Substances 0.000 description 2
- 229910052751 metal Inorganic materials 0.000 description 2
- 239000002184 metal Substances 0.000 description 2
- 150000002739 metals Chemical class 0.000 description 2
- 229930182817 methionine Natural products 0.000 description 2
- 229920001542 oligosaccharide Polymers 0.000 description 2
- 150000002482 oligosaccharides Chemical class 0.000 description 2
- 230000036961 partial effect Effects 0.000 description 2
- 230000026731 phosphorylation Effects 0.000 description 2
- 238000006366 phosphorylation reaction Methods 0.000 description 2
- 239000011148 porous material Substances 0.000 description 2
- 230000008569 process Effects 0.000 description 2
- 230000004952 protein activity Effects 0.000 description 2
- ZCCUUQDIBDJBTK-UHFFFAOYSA-N psoralen Chemical compound C1=C2OC(=O)C=CC2=CC2=C1OC=C2 ZCCUUQDIBDJBTK-UHFFFAOYSA-N 0.000 description 2
- 238000000746 purification Methods 0.000 description 2
- AWUCVROLDVIAJX-GSVOUGTGSA-N sn-glycerol 3-phosphate Chemical compound OC[C@@H](O)COP(O)(O)=O AWUCVROLDVIAJX-GSVOUGTGSA-N 0.000 description 2
- 238000013519 translation Methods 0.000 description 2
- LENZDBCJOHFCAS-UHFFFAOYSA-N tris Chemical compound OCC(N)(CO)CO LENZDBCJOHFCAS-UHFFFAOYSA-N 0.000 description 2
- 102000006670 unclassified proteins Human genes 0.000 description 2
- 108020004732 unclassified proteins Proteins 0.000 description 2
- 238000011144 upstream manufacturing Methods 0.000 description 2
- 230000009105 vegetative growth Effects 0.000 description 2
- 238000011179 visual inspection Methods 0.000 description 2
- 150000003741 xylose derivatives Chemical class 0.000 description 2
- 230000004127 xylose metabolism Effects 0.000 description 2
- LGQKSQQRKHFMLI-SJYYZXOBSA-N (2s,3r,4s,5r)-2-[(3r,4r,5r,6r)-4,5,6-trihydroxyoxan-3-yl]oxyoxane-3,4,5-triol Chemical compound O[C@@H]1[C@@H](O)[C@H](O)CO[C@H]1O[C@H]1[C@H](O)[C@@H](O)[C@H](O)OC1 LGQKSQQRKHFMLI-SJYYZXOBSA-N 0.000 description 1
- AAWZDTNXLSGCEK-LNVDRNJUSA-N (3r,5r)-1,3,4,5-tetrahydroxycyclohexane-1-carboxylic acid Chemical compound O[C@@H]1CC(O)(C(O)=O)C[C@@H](O)C1O AAWZDTNXLSGCEK-LNVDRNJUSA-N 0.000 description 1
- ASJSAQIRZKANQN-CRCLSJGQSA-N 2-deoxy-D-ribose Chemical compound OC[C@@H](O)[C@@H](O)CC=O ASJSAQIRZKANQN-CRCLSJGQSA-N 0.000 description 1
- VXGRJERITKFWPL-UHFFFAOYSA-N 4',5'-Dihydropsoralen Natural products C1=C2OC(=O)C=CC2=CC2=C1OCC2 VXGRJERITKFWPL-UHFFFAOYSA-N 0.000 description 1
- LGQKSQQRKHFMLI-UHFFFAOYSA-N 4-O-beta-D-xylopyranosyl-beta-D-xylopyranose Natural products OC1C(O)C(O)COC1OC1C(O)C(O)C(O)OC1 LGQKSQQRKHFMLI-UHFFFAOYSA-N 0.000 description 1
- 108010034869 6-phospho-beta-glucosidase Proteins 0.000 description 1
- 241001019659 Acremonium <Plectosphaerellaceae> Species 0.000 description 1
- 108010085238 Actins Proteins 0.000 description 1
- 108700028369 Alleles Proteins 0.000 description 1
- 239000004475 Arginine Substances 0.000 description 1
- 241000235349 Ascomycota Species 0.000 description 1
- DCXYFEDJOCDNAF-UHFFFAOYSA-N Asparagine Natural products OC(=O)C(N)CC(N)=O DCXYFEDJOCDNAF-UHFFFAOYSA-N 0.000 description 1
- 241001513093 Aspergillus awamori Species 0.000 description 1
- 241000892910 Aspergillus foetidus Species 0.000 description 1
- 241001480052 Aspergillus japonicus Species 0.000 description 1
- 241000351920 Aspergillus nidulans Species 0.000 description 1
- 241000228245 Aspergillus niger Species 0.000 description 1
- 235000002247 Aspergillus oryzae Nutrition 0.000 description 1
- 241000589151 Azotobacter Species 0.000 description 1
- 241000304886 Bacilli Species 0.000 description 1
- 241000193830 Bacillus <bacterium> Species 0.000 description 1
- 241001330002 Bambuseae Species 0.000 description 1
- 241001135755 Betaproteobacteria Species 0.000 description 1
- 241001608472 Bifidobacterium longum Species 0.000 description 1
- ZOXJGFHDIHLPTG-UHFFFAOYSA-N Boron Chemical compound [B] ZOXJGFHDIHLPTG-UHFFFAOYSA-N 0.000 description 1
- 241000123649 Botryotinia Species 0.000 description 1
- 241000123650 Botrytis cinerea Species 0.000 description 1
- 244000027711 Brettanomyces bruxellensis Species 0.000 description 1
- 235000000287 Brettanomyces bruxellensis Nutrition 0.000 description 1
- 241000776348 Carboxydocella Species 0.000 description 1
- 229920002101 Chitin Polymers 0.000 description 1
- 229920001661 Chitosan Polymers 0.000 description 1
- 241000233652 Chytridiomycota Species 0.000 description 1
- 241001508813 Clavispora lusitaniae Species 0.000 description 1
- 241000193403 Clostridium Species 0.000 description 1
- 241000228437 Cochliobolus Species 0.000 description 1
- 108091026890 Coding region Proteins 0.000 description 1
- 108020004705 Codon Proteins 0.000 description 1
- AAWZDTNXLSGCEK-UHFFFAOYSA-N Cordycepinsaeure Natural products OC1CC(O)(C(O)=O)CC(O)C1O AAWZDTNXLSGCEK-UHFFFAOYSA-N 0.000 description 1
- 241000186226 Corynebacterium glutamicum Species 0.000 description 1
- 108020003264 Cotransporters Proteins 0.000 description 1
- 102000034534 Cotransporters Human genes 0.000 description 1
- 241000192700 Cyanobacteria Species 0.000 description 1
- WQZGKKKJIJFFOK-QTVWNMPRSA-N D-mannopyranose Chemical compound OC[C@H]1OC(O)[C@@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-QTVWNMPRSA-N 0.000 description 1
- SRBFZHDQGSBBOR-SOOFDHNKSA-N D-ribopyranose Chemical compound O[C@@H]1COC(O)[C@H](O)[C@@H]1O SRBFZHDQGSBBOR-SOOFDHNKSA-N 0.000 description 1
- ZAQJHHRNXZUBTE-UHFFFAOYSA-N D-threo-2-Pentulose Natural products OCC(O)C(O)C(=O)CO ZAQJHHRNXZUBTE-UHFFFAOYSA-N 0.000 description 1
- SQNRKWHRVIAKLP-UHFFFAOYSA-N D-xylobiose Natural products O=CC(O)C(O)C(CO)OC1OCC(O)C(O)C1O SQNRKWHRVIAKLP-UHFFFAOYSA-N 0.000 description 1
- 102000012410 DNA Ligases Human genes 0.000 description 1
- 108010061982 DNA Ligases Proteins 0.000 description 1
- 230000004544 DNA amplification Effects 0.000 description 1
- 102000016928 DNA-directed DNA polymerase Human genes 0.000 description 1
- 108010014303 DNA-directed DNA polymerase Proteins 0.000 description 1
- 102000004163 DNA-directed RNA polymerases Human genes 0.000 description 1
- 108090000626 DNA-directed RNA polymerases Proteins 0.000 description 1
- 239000004375 Dextrin Substances 0.000 description 1
- 229920001353 Dextrin Polymers 0.000 description 1
- 241000588700 Dickeya chrysanthemi Species 0.000 description 1
- 244000182625 Dictamnus albus Species 0.000 description 1
- 101710121765 Endo-1,4-beta-xylanase Proteins 0.000 description 1
- 241000178951 Endomyces Species 0.000 description 1
- 241000588914 Enterobacter Species 0.000 description 1
- 241000588921 Enterobacteriaceae Species 0.000 description 1
- 241000588698 Erwinia Species 0.000 description 1
- 241000701959 Escherichia virus Lambda Species 0.000 description 1
- 108050001049 Extracellular proteins Proteins 0.000 description 1
- 229930091371 Fructose Natural products 0.000 description 1
- RFSUNEUAIZKAJO-ARQDHWQXSA-N Fructose Chemical compound OC[C@H]1O[C@](O)(CO)[C@@H](O)[C@@H]1O RFSUNEUAIZKAJO-ARQDHWQXSA-N 0.000 description 1
- 239000005715 Fructose Substances 0.000 description 1
- 241000145614 Fusarium bactridioides Species 0.000 description 1
- 241000223194 Fusarium culmorum Species 0.000 description 1
- 241000223195 Fusarium graminearum Species 0.000 description 1
- 241001014439 Fusarium sarcochroum Species 0.000 description 1
- 241000223192 Fusarium sporotrichioides Species 0.000 description 1
- 241001465753 Fusarium torulosum Species 0.000 description 1
- 241000567178 Fusarium venenatum Species 0.000 description 1
- 206010071602 Genetic polymorphism Diseases 0.000 description 1
- 101100218337 Gibberella zeae (strain ATCC MYA-4620 / CBS 123657 / FGSC 9075 / NRRL 31084 / PH-1) aurL2 gene Proteins 0.000 description 1
- 229920001503 Glucan Polymers 0.000 description 1
- 108010073178 Glucan 1,4-alpha-Glucosidase Proteins 0.000 description 1
- 102100022624 Glucoamylase Human genes 0.000 description 1
- 241000238631 Hexapoda Species 0.000 description 1
- 241000223198 Humicola Species 0.000 description 1
- 241001480714 Humicola insolens Species 0.000 description 1
- 108010044467 Isoenzymes Proteins 0.000 description 1
- 241000588748 Klebsiella Species 0.000 description 1
- QNAYBMKLOCPYGJ-REOHCLBHSA-N L-alanine Chemical compound C[C@H](N)C(O)=O QNAYBMKLOCPYGJ-REOHCLBHSA-N 0.000 description 1
- DCXYFEDJOCDNAF-REOHCLBHSA-N L-asparagine Chemical compound OC(=O)[C@@H](N)CC(N)=O DCXYFEDJOCDNAF-REOHCLBHSA-N 0.000 description 1
- AGPKZVBTJJNPAG-WHFBIAKZSA-N L-isoleucine Chemical compound CC[C@H](C)[C@H](N)C(O)=O AGPKZVBTJJNPAG-WHFBIAKZSA-N 0.000 description 1
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 1
- COLNVLDHVKWLRT-QMMMGPOBSA-N L-phenylalanine Chemical compound OC(=O)[C@@H](N)CC1=CC=CC=C1 COLNVLDHVKWLRT-QMMMGPOBSA-N 0.000 description 1
- SHZGCJCMOBCMKK-JFNONXLTSA-N L-rhamnopyranose Chemical compound C[C@@H]1OC(O)[C@H](O)[C@H](O)[C@H]1O SHZGCJCMOBCMKK-JFNONXLTSA-N 0.000 description 1
- OUYCCCASQSFEME-QMMMGPOBSA-N L-tyrosine Chemical compound OC(=O)[C@@H](N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-QMMMGPOBSA-N 0.000 description 1
- KZSNJWFQEVHDMF-BYPYZUCNSA-N L-valine Chemical compound CC(C)[C@H](N)C(O)=O KZSNJWFQEVHDMF-BYPYZUCNSA-N 0.000 description 1
- ZAQJHHRNXZUBTE-WVZVXSGGSA-N L-xylulose Chemical compound OC[C@H](O)[C@@H](O)C(=O)CO ZAQJHHRNXZUBTE-WVZVXSGGSA-N 0.000 description 1
- 101150017281 LAC2 gene Proteins 0.000 description 1
- 241001149420 Laccaria bicolor Species 0.000 description 1
- 241000235087 Lachancea kluyveri Species 0.000 description 1
- 241000186610 Lactobacillus sp. Species 0.000 description 1
- 241001494496 Leersia Species 0.000 description 1
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 description 1
- 101710123588 Low-affinity glucose transporter Proteins 0.000 description 1
- 239000004472 Lysine Substances 0.000 description 1
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 description 1
- 229920000057 Mannan Polymers 0.000 description 1
- 238000007476 Maximum Likelihood Methods 0.000 description 1
- 102000005455 Monosaccharide Transport Proteins Human genes 0.000 description 1
- 108010006769 Monosaccharide Transport Proteins Proteins 0.000 description 1
- 101710132674 Monosaccharide transporter Proteins 0.000 description 1
- 241000235395 Mucor Species 0.000 description 1
- 241000226677 Myceliophthora Species 0.000 description 1
- 241001674208 Mycothermus thermophilus Species 0.000 description 1
- 229930182474 N-glycoside Natural products 0.000 description 1
- 241001226034 Nectria <echinoderm> Species 0.000 description 1
- 241001507755 Neosartorya Species 0.000 description 1
- 241001477116 Neurospora crassa OR74A Species 0.000 description 1
- 101710163270 Nuclease Proteins 0.000 description 1
- 108010038807 Oligopeptides Proteins 0.000 description 1
- 102000015636 Oligopeptides Human genes 0.000 description 1
- 101100449758 Onchocerca volvulus GST1 gene Proteins 0.000 description 1
- 241000191998 Pediococcus acidilactici Species 0.000 description 1
- 241000222393 Phanerochaete chrysosporium Species 0.000 description 1
- 241000425347 Phyla <beetle> Species 0.000 description 1
- 241001599925 Polaromonas naphthalenivorans Species 0.000 description 1
- 241001492489 Postia placenta Species 0.000 description 1
- 108010029485 Protein Isoforms Proteins 0.000 description 1
- 102000001708 Protein Isoforms Human genes 0.000 description 1
- 108010076504 Protein Sorting Signals Proteins 0.000 description 1
- 241000588769 Proteus <enterobacteria> Species 0.000 description 1
- 241000589516 Pseudomonas Species 0.000 description 1
- KDCGOANMDULRCW-UHFFFAOYSA-N Purine Natural products N1=CNC2=NC=NC2=C1 KDCGOANMDULRCW-UHFFFAOYSA-N 0.000 description 1
- 241000228453 Pyrenophora Species 0.000 description 1
- CZPWVGJYEJSRLH-UHFFFAOYSA-N Pyrimidine Chemical compound C1=CN=CN=C1 CZPWVGJYEJSRLH-UHFFFAOYSA-N 0.000 description 1
- LCTONWCANYUPML-UHFFFAOYSA-M Pyruvate Chemical compound CC(=O)C([O-])=O LCTONWCANYUPML-UHFFFAOYSA-M 0.000 description 1
- AAWZDTNXLSGCEK-ZHQZDSKASA-N Quinic acid Natural products O[C@H]1CC(O)(C(O)=O)C[C@H](O)C1O AAWZDTNXLSGCEK-ZHQZDSKASA-N 0.000 description 1
- 241000235403 Rhizomucor miehei Species 0.000 description 1
- 241000191023 Rhodobacter capsulatus Species 0.000 description 1
- 108091028664 Ribonucleotide Proteins 0.000 description 1
- 101100376256 Saccharomyces cerevisiae (strain ATCC 204508 / S288c) YHR210C gene Proteins 0.000 description 1
- 101100432594 Saccharomyces cerevisiae (strain ATCC 204508 / S288c) YNR071C gene Proteins 0.000 description 1
- 235000001006 Saccharomyces cerevisiae var diastaticus Nutrition 0.000 description 1
- 244000206963 Saccharomyces cerevisiae var. diastaticus Species 0.000 description 1
- 241000204893 Saccharomyces douglasii Species 0.000 description 1
- 241000607142 Salmonella Species 0.000 description 1
- 241000235346 Schizosaccharomyces Species 0.000 description 1
- 241000221662 Sclerotinia Species 0.000 description 1
- 241000223255 Scytalidium Species 0.000 description 1
- 238000012300 Sequence Analysis Methods 0.000 description 1
- MTCFGRXMJLQNBG-UHFFFAOYSA-N Serine Natural products OCC(N)C(O)=O MTCFGRXMJLQNBG-UHFFFAOYSA-N 0.000 description 1
- 241000607720 Serratia Species 0.000 description 1
- 241000607768 Shigella Species 0.000 description 1
- 108091081024 Start codon Proteins 0.000 description 1
- 244000057717 Streptococcus lactis Species 0.000 description 1
- 235000014897 Streptococcus lactis Nutrition 0.000 description 1
- 241000192707 Synechococcus Species 0.000 description 1
- 241000192584 Synechocystis Species 0.000 description 1
- 241001540751 Talaromyces ruber Species 0.000 description 1
- 241000223258 Thermomyces lanuginosus Species 0.000 description 1
- 241001494489 Thielavia Species 0.000 description 1
- 241001495429 Thielavia terrestris Species 0.000 description 1
- RYYWUUFWQRZTIU-UHFFFAOYSA-N Thiophosphoric acid Chemical class OP(O)(S)=O RYYWUUFWQRZTIU-UHFFFAOYSA-N 0.000 description 1
- AYFVYJQAPQTCCC-UHFFFAOYSA-N Threonine Natural products CC(O)C(N)C(O)=O AYFVYJQAPQTCCC-UHFFFAOYSA-N 0.000 description 1
- 239000004473 Threonine Substances 0.000 description 1
- 241001149964 Tolypocladium Species 0.000 description 1
- 241000223259 Trichoderma Species 0.000 description 1
- 241000223260 Trichoderma harzianum Species 0.000 description 1
- 241000378866 Trichoderma koningii Species 0.000 description 1
- 241000223262 Trichoderma longibrachiatum Species 0.000 description 1
- 241000223261 Trichoderma viride Species 0.000 description 1
- 244000223977 Tuber melanosporum Species 0.000 description 1
- KZSNJWFQEVHDMF-UHFFFAOYSA-N Valine Natural products CC(C)C(N)C(O)=O KZSNJWFQEVHDMF-UHFFFAOYSA-N 0.000 description 1
- 241000700605 Viruses Species 0.000 description 1
- 241000863000 Vitreoscilla Species 0.000 description 1
- 101150101877 XI gene Proteins 0.000 description 1
- 241000589636 Xanthomonas campestris Species 0.000 description 1
- 241000235013 Yarrowia Species 0.000 description 1
- 241000235015 Yarrowia lipolytica Species 0.000 description 1
- HCHKCACWOHOZIP-UHFFFAOYSA-N Zinc Chemical compound [Zn] HCHKCACWOHOZIP-UHFFFAOYSA-N 0.000 description 1
- 241000758405 Zoopagomycotina Species 0.000 description 1
- 241000235017 Zygosaccharomyces Species 0.000 description 1
- 241001231403 [Nectria] haematococca Species 0.000 description 1
- 238000002679 ablation Methods 0.000 description 1
- 230000002159 abnormal effect Effects 0.000 description 1
- 230000005856 abnormality Effects 0.000 description 1
- 230000002411 adverse Effects 0.000 description 1
- 235000004279 alanine Nutrition 0.000 description 1
- 238000012867 alanine scanning Methods 0.000 description 1
- 150000001298 alcohols Chemical class 0.000 description 1
- 125000000217 alkyl group Chemical group 0.000 description 1
- 210000004102 animal cell Anatomy 0.000 description 1
- 238000000137 annealing Methods 0.000 description 1
- 238000013459 approach Methods 0.000 description 1
- ODKSFYDXXFIFQN-UHFFFAOYSA-N arginine Natural products OC(=O)C(N)CCCNC(N)=N ODKSFYDXXFIFQN-UHFFFAOYSA-N 0.000 description 1
- 235000009582 asparagine Nutrition 0.000 description 1
- 229960001230 asparagine Drugs 0.000 description 1
- QVGXLLKOCUKJST-UHFFFAOYSA-N atomic oxygen Chemical compound [O] QVGXLLKOCUKJST-UHFFFAOYSA-N 0.000 description 1
- 238000007630 basic procedure Methods 0.000 description 1
- 229940009291 bifidobacterium longum Drugs 0.000 description 1
- 230000036983 biotransformation Effects 0.000 description 1
- 229910052796 boron Inorganic materials 0.000 description 1
- 150000004657 carbamic acid derivatives Chemical class 0.000 description 1
- 150000001720 carbohydrates Chemical class 0.000 description 1
- 239000001768 carboxy methyl cellulose Substances 0.000 description 1
- 235000010948 carboxy methyl cellulose Nutrition 0.000 description 1
- 239000008112 carboxymethyl-cellulose Substances 0.000 description 1
- 230000034303 cell budding Effects 0.000 description 1
- 230000008618 cell wall macromolecule catabolic process Effects 0.000 description 1
- 230000001413 cellular effect Effects 0.000 description 1
- 210000003850 cellular structure Anatomy 0.000 description 1
- 239000002738 chelating agent Substances 0.000 description 1
- 238000012824 chemical production Methods 0.000 description 1
- 238000007621 cluster analysis Methods 0.000 description 1
- 238000011109 contamination Methods 0.000 description 1
- 230000002596 correlated effect Effects 0.000 description 1
- 239000012228 culture supernatant Substances 0.000 description 1
- 235000018417 cysteine Nutrition 0.000 description 1
- XUJNEKJLAYXESH-UHFFFAOYSA-N cysteine Natural products SCC(N)C(O)=O XUJNEKJLAYXESH-UHFFFAOYSA-N 0.000 description 1
- 230000001461 cytolytic effect Effects 0.000 description 1
- 230000001086 cytosolic effect Effects 0.000 description 1
- 238000000354 decomposition reaction Methods 0.000 description 1
- 230000002950 deficient Effects 0.000 description 1
- 239000007857 degradation product Substances 0.000 description 1
- 230000000593 degrading effect Effects 0.000 description 1
- 235000019425 dextrin Nutrition 0.000 description 1
- 238000010586 diagram Methods 0.000 description 1
- 125000000600 disaccharide group Chemical group 0.000 description 1
- 150000002016 disaccharides Chemical class 0.000 description 1
- NAGJZTKCGNOGPW-UHFFFAOYSA-N dithiophosphoric acid Chemical class OP(O)(S)=S NAGJZTKCGNOGPW-UHFFFAOYSA-N 0.000 description 1
- 230000003828 downregulation Effects 0.000 description 1
- 238000001962 electrophoresis Methods 0.000 description 1
- 238000013401 experimental design Methods 0.000 description 1
- 239000002803 fossil fuel Substances 0.000 description 1
- 238000010230 functional analysis Methods 0.000 description 1
- 108020001507 fusion proteins Proteins 0.000 description 1
- 102000037865 fusion proteins Human genes 0.000 description 1
- 101150054547 galM gene Proteins 0.000 description 1
- 230000030279 gene silencing Effects 0.000 description 1
- 238000003167 genetic complementation Methods 0.000 description 1
- 230000006377 glucose transport Effects 0.000 description 1
- ZDXPYRJPNDTMRX-UHFFFAOYSA-N glutamine Natural products OC(=O)C(N)CCC(N)=O ZDXPYRJPNDTMRX-UHFFFAOYSA-N 0.000 description 1
- 150000002402 hexoses Chemical group 0.000 description 1
- 238000004128 high performance liquid chromatography Methods 0.000 description 1
- 238000002744 homologous recombination Methods 0.000 description 1
- 230000006801 homologous recombination Effects 0.000 description 1
- 230000002779 inactivation Effects 0.000 description 1
- 239000000411 inducer Substances 0.000 description 1
- 238000009655 industrial fermentation Methods 0.000 description 1
- 239000003112 inhibitor Substances 0.000 description 1
- 238000003780 insertion Methods 0.000 description 1
- 230000037431 insertion Effects 0.000 description 1
- 230000010354 integration Effects 0.000 description 1
- 239000013067 intermediate product Substances 0.000 description 1
- 238000011835 investigation Methods 0.000 description 1
- 150000002500 ions Chemical class 0.000 description 1
- 229960000310 isoleucine Drugs 0.000 description 1
- AGPKZVBTJJNPAG-UHFFFAOYSA-N isoleucine Natural products CCC(C)C(N)C(O)=O AGPKZVBTJJNPAG-UHFFFAOYSA-N 0.000 description 1
- 239000004310 lactic acid Substances 0.000 description 1
- 235000014655 lactic acid Nutrition 0.000 description 1
- 239000012978 lignocellulosic material Substances 0.000 description 1
- 239000002502 liposome Substances 0.000 description 1
- 230000033001 locomotion Effects 0.000 description 1
- 229920002521 macromolecule Polymers 0.000 description 1
- 238000004949 mass spectrometry Methods 0.000 description 1
- 239000012092 media component Substances 0.000 description 1
- 230000001404 mediated effect Effects 0.000 description 1
- HEBKCHPVOIAQTA-UHFFFAOYSA-N meso ribitol Natural products OCC(O)C(O)C(O)CO HEBKCHPVOIAQTA-UHFFFAOYSA-N 0.000 description 1
- 230000004060 metabolic process Effects 0.000 description 1
- YACKEPLHDIMKIO-UHFFFAOYSA-N methylphosphonic acid Chemical class CP(O)(O)=O YACKEPLHDIMKIO-UHFFFAOYSA-N 0.000 description 1
- 230000000813 microbial effect Effects 0.000 description 1
- 230000005012 migration Effects 0.000 description 1
- 238000013508 migration Methods 0.000 description 1
- 108091005573 modified proteins Proteins 0.000 description 1
- 102000035118 modified proteins Human genes 0.000 description 1
- 238000002703 mutagenesis Methods 0.000 description 1
- 231100000350 mutagenesis Toxicity 0.000 description 1
- 229960003966 nicotinamide Drugs 0.000 description 1
- 235000005152 nicotinamide Nutrition 0.000 description 1
- 239000011570 nicotinamide Substances 0.000 description 1
- 230000000269 nucleophilic effect Effects 0.000 description 1
- 235000016709 nutrition Nutrition 0.000 description 1
- 238000012261 overproduction Methods 0.000 description 1
- 230000001590 oxidative effect Effects 0.000 description 1
- 239000001301 oxygen Substances 0.000 description 1
- 229910052760 oxygen Inorganic materials 0.000 description 1
- 239000003973 paint Substances 0.000 description 1
- COLNVLDHVKWLRT-UHFFFAOYSA-N phenylalanine Natural products OC(=O)C(N)CC1=CC=CC=C1 COLNVLDHVKWLRT-UHFFFAOYSA-N 0.000 description 1
- 150000008298 phosphoramidates Chemical class 0.000 description 1
- 150000008300 phosphoramidites Chemical class 0.000 description 1
- OJMIONKXNSYLSR-UHFFFAOYSA-N phosphorous acid Chemical class OP(O)O OJMIONKXNSYLSR-UHFFFAOYSA-N 0.000 description 1
- 238000013081 phylogenetic analysis Methods 0.000 description 1
- 229920000729 poly(L-lysine) polymer Polymers 0.000 description 1
- 229920001282 polysaccharide Polymers 0.000 description 1
- 239000005017 polysaccharide Substances 0.000 description 1
- 150000004804 polysaccharides Chemical class 0.000 description 1
- 239000002243 precursor Substances 0.000 description 1
- 125000002924 primary amino group Chemical group [H]N([H])* 0.000 description 1
- 210000001236 prokaryotic cell Anatomy 0.000 description 1
- 210000004777 protein coat Anatomy 0.000 description 1
- 238000001243 protein synthesis Methods 0.000 description 1
- IGFXRKMLLMBKSA-UHFFFAOYSA-N purine Chemical compound N1=C[N]C2=NC=NC2=C1 IGFXRKMLLMBKSA-UHFFFAOYSA-N 0.000 description 1
- 238000011002 quantification Methods 0.000 description 1
- 230000002285 radioactive effect Effects 0.000 description 1
- 238000003259 recombinant expression Methods 0.000 description 1
- 238000006479 redox reaction Methods 0.000 description 1
- 238000011160 research Methods 0.000 description 1
- 239000011347 resin Substances 0.000 description 1
- 229920005989 resin Polymers 0.000 description 1
- 230000004044 response Effects 0.000 description 1
- 238000003757 reverse transcription PCR Methods 0.000 description 1
- 230000002441 reversible effect Effects 0.000 description 1
- 239000002336 ribonucleotide Substances 0.000 description 1
- 125000002652 ribonucleotide group Chemical group 0.000 description 1
- 239000000523 sample Substances 0.000 description 1
- 229930000044 secondary metabolite Natural products 0.000 description 1
- 230000035945 sensitivity Effects 0.000 description 1
- 239000013605 shuttle vector Substances 0.000 description 1
- 235000021309 simple sugar Nutrition 0.000 description 1
- 150000003384 small molecules Chemical class 0.000 description 1
- 238000007619 statistical method Methods 0.000 description 1
- 239000000126 substance Substances 0.000 description 1
- 239000006228 supernatant Substances 0.000 description 1
- 239000000725 suspension Substances 0.000 description 1
- 230000002195 synergetic effect Effects 0.000 description 1
- 238000004885 tandem mass spectrometry Methods 0.000 description 1
- 239000003053 toxin Substances 0.000 description 1
- 231100000765 toxin Toxicity 0.000 description 1
- 108700012359 toxins Proteins 0.000 description 1
- 238000010361 transduction Methods 0.000 description 1
- 230000026683 transduction Effects 0.000 description 1
- OUYCCCASQSFEME-UHFFFAOYSA-N tyrosine Natural products OC(=O)C(N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-UHFFFAOYSA-N 0.000 description 1
- 230000003827 upregulation Effects 0.000 description 1
- 239000004474 valine Substances 0.000 description 1
- 230000035899 viability Effects 0.000 description 1
- 229920001221 xylan Polymers 0.000 description 1
- 150000004823 xylans Chemical class 0.000 description 1
- 229910052725 zinc Inorganic materials 0.000 description 1
- 239000011701 zinc Substances 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P21/00—Preparation of peptides or proteins
- C12P21/02—Preparation of peptides or proteins having a known sequence of two or more amino acids, e.g. glutathione
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/37—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from fungi
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P7/00—Preparation of oxygen-containing organic compounds
- C12P7/02—Preparation of oxygen-containing organic compounds containing a hydroxy group
- C12P7/04—Preparation of oxygen-containing organic compounds containing a hydroxy group acyclic
- C12P7/06—Ethanol, i.e. non-beverage
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/705—Receptors; Cell surface antigens; Cell surface determinants
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N1/00—Microorganisms, e.g. protozoa; Compositions thereof; Processes of propagating, maintaining or preserving microorganisms or compositions thereof; Processes of preparing or isolating a composition containing a microorganism; Culture media therefor
- C12N1/14—Fungi; Culture media therefor
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/52—Genes encoding for enzymes or proenzymes
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/80—Vectors or expression systems specially adapted for eukaryotic hosts for fungi
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/80—Vectors or expression systems specially adapted for eukaryotic hosts for fungi
- C12N15/81—Vectors or expression systems specially adapted for eukaryotic hosts for fungi for yeasts
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/87—Introduction of foreign genetic material using processes not otherwise provided for, e.g. co-transformation
-
- Y—GENERAL TAGGING OF NEW TECHNOLOGICAL DEVELOPMENTS; GENERAL TAGGING OF CROSS-SECTIONAL TECHNOLOGIES SPANNING OVER SEVERAL SECTIONS OF THE IPC; TECHNICAL SUBJECTS COVERED BY FORMER USPC CROSS-REFERENCE ART COLLECTIONS [XRACs] AND DIGESTS
- Y02—TECHNOLOGIES OR APPLICATIONS FOR MITIGATION OR ADAPTATION AGAINST CLIMATE CHANGE
- Y02E—REDUCTION OF GREENHOUSE GAS [GHG] EMISSIONS, RELATED TO ENERGY GENERATION, TRANSMISSION OR DISTRIBUTION
- Y02E50/00—Technologies for the production of fuel of non-fossil origin
- Y02E50/10—Biofuels, e.g. bio-diesel
Landscapes
- Life Sciences & Earth Sciences (AREA)
- Health & Medical Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Genetics & Genomics (AREA)
- Organic Chemistry (AREA)
- Engineering & Computer Science (AREA)
- Zoology (AREA)
- Wood Science & Technology (AREA)
- Biotechnology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- General Engineering & Computer Science (AREA)
- Biomedical Technology (AREA)
- Biochemistry (AREA)
- General Health & Medical Sciences (AREA)
- Molecular Biology (AREA)
- Biophysics (AREA)
- Microbiology (AREA)
- Mycology (AREA)
- Plant Pathology (AREA)
- Physics & Mathematics (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Medicinal Chemistry (AREA)
- Gastroenterology & Hepatology (AREA)
- General Chemical & Material Sciences (AREA)
- Chemical Kinetics & Catalysis (AREA)
- Cell Biology (AREA)
- Immunology (AREA)
- Toxicology (AREA)
- Virology (AREA)
- Tropical Medicine & Parasitology (AREA)
- Botany (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Preparation Of Compounds By Using Micro-Organisms (AREA)
- Enzymes And Modification Thereof (AREA)
- Peptides Or Proteins (AREA)
Abstract
本開示は、ポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞に関し、ここで、当該ポリペプチドは当該細胞にセロデキストリンを輸送する。本開示は、セロデキストリンを含んでいる培地における細胞の成長を促進させる方法、セルロースから得られた糖およびヘミセルロースから得られた糖を共発酵させる方法、ならびにポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備することによって、炭化水素または炭化水素の誘導体を製造する方法にさらに関し、ここで、当該ポリペプチドは当該細胞にセロデキストリンを輸送する。本開示は、ポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞、細胞へのペントースの輸送を促進させる方法、ペントース糖を含んでいる培地における細胞の成長を向上させる方法、およびポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備することによって、炭化水素または炭化水素の誘導体を製造する方法に関し、ここで、当該ポリペプチドは当該細胞にペントースを輸送する。
Description
〔関連出願の相互参照〕
本願は、参照によってそれらの全体が本明細書に援用される米国仮出願第61/285,526号(出願日:2009年12月10日)および米国仮出願第61/271,833号(出願日:2009年7月24日)の利益を主張する。
本願は、参照によってそれらの全体が本明細書に援用される米国仮出願第61/285,526号(出願日:2009年12月10日)および米国仮出願第61/271,833号(出願日:2009年7月24日)の利益を主張する。
本開示は、細胞への糖の輸送を促進させる方法および組成物、細胞の成長を促進させる方法および組成物、炭化水素および炭化水素の誘導体の合成を促進させる方法および組成物、ならびにセルロース由来の糖およびヘミセルロース由来の糖を共発酵させる方法および組成物に関する。
生物燃料は、エネルギーの安全保障、資源の継続的維持および世界的な気候変動に対する関心の増大のために、集中的に研究されている(Lynd et al., 1991)。植物由来のリグノセルロース性材料の、燃料への生物変換は、化石燃料の化学的生成に変わる魅力的な代替物と見做されている(Lynd et al. 2008;Hahn-Hagerdal et al. 2006)。
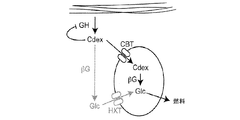
エタノールへのリグノセルロース性のバイオマスの変換を、効率的に実施するための微生物の改変は、依然として生物燃料の分野の主な目標である。多大な探索は、単純な糖をアルコールへ天然に発酵させる微生物を遺伝的に改変して、1つの細胞内におけるリグノセルロース性のバイオマス重合体の分解およびエタノール産生を微生物にとって可能にさせるセルラーゼおよび他の酵素を発現させることに焦点が当てられている。しかし、十分に研究されていない領域は糖の輸送である。糖の取込み能を向上させるための糖輸送の制御の理解および微生物の遺伝的な改変は、効率を非常に向上させる(Stephanopoulos 2007)。さらに、セルラーゼ発現およびセルラーゼ活性の制御に関与する他の種類のタンパク質は、依然として十分に調査されるべき状態にある。
Saccharomyces cerevisiae(パン酵母としても知られている)は、エタノールへのヘキソース糖の生物変換のために、数千年間にわたって使用されている。また、Saccharomyces cerevisiaeは、エタノールへのD−グルコースの大規模な工業的発酵のために最も広く使用されている微生物である。S. cerevisiaeは、生物燃料へのリグノセルロース性のバイオマスの生物変換にとって最も好適な候補物である(van Maris et al., 2006)。S. cerevisiaeは、十分に研究されている遺伝的背景および生理学的背景、豊富な遺伝学的な手段、ならびにリグノセルロース性の加水分解物に見られる阻害物質および高濃度のエタノールに対する高い耐性を有している(Jeffries 2006)。また、低pHにおけるS. cerevisiaeの発酵によって、発酵の間における細菌汚染が防止され得る。
残念ながら、野生型のS. cerevisiaeはペントース糖を利用できない(Hector et al., 2008)。この制限を解消させるために、ペントースを消化する生物に由来するペントース利用経路がS. cerevisiaeに導入されており、D−キシロースおよびL−アラビノースの発酵を可能にしている(Hahn-Hagerdal et al., 2007;Brat et al., 2009; Wisselink et al., 2007, 2009;Wiedemann and Boles 2008;Karhumma et al., 2006)。しかし、生物燃料へのペントース糖の効率的な変換は、複数の問題(細胞性の酸化還元反応の不均衡、ペントースリン酸化経路の低い作用、および細胞への効率的なペントース輸送の欠如が挙げられる)によって制限されている(Hector et al., 2008)。
さらに、天然の微生物および改変微生物は、グルコース発酵と比べて、キシロース発酵の間におけるエタノール許容性の低下を示す(Jeffries and Jin 2000)。より低い発酵速度と相まって、キシロース発酵の間におけるエタノール許容性の低下は、セルロース性の加水分解物に存在している高濃度のグルコース(70〜100g/L未満)およびキシロース(40〜60g/L未満)を含有している糖の混合物の発酵において重大な問題をもたらす。微生物はグルコースを優先的に利用するので、グルコースの枯渇時(細胞がキシロースを使用し始めるとき)に、すでにエタノール濃度はキシロースの発酵速度をさらに低下させるほど十分に高い。結果として、“グルコース抑制”のために、グルコース枯渇後に続いて起こるキシロースの利用は、セルロース性の加水分解物における混合糖を首尾よく利用するために解消されるべき重要な課題である。
したがって、リグノセルロースに基づく向上された成長およびリグノセルロース分解から生じる化合物の向上された取込みを目的として、リグノセルロースの分解および微生物の改変における利用に重要なさらなる遺伝子の同定に対する必要性がある。
これらの必要性を満たすために、本明細書に記載の本発明は、細胞へのセロデキストリンの輸送を促進させる方法、セロデキストリンを含有している培地における細胞の成長を促進させる方法、セルロース由来の糖およびヘミセルロース由来の糖を共発酵させる方法、ならびにセロデキストリンを細胞に輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備することによって、炭化水素または炭化水素の誘導体を製造する方法を提供する。本明細書にさらに記載されているのは、セロデキストリンを細胞に輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞である。本明細書にさらに記載されているのは、ペントースを細胞に輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞、細胞へのペントースの輸送を促進させる方法、ペントース糖を含有している培地における細胞の成長を促進させる方法、ならびにペントースを細胞に輸送するポリペプチドをコードしているポリヌクレオチドを含んでいる宿主細胞を準備することによって、炭化水素または炭化水素の誘導体を製造する方法を提供する。
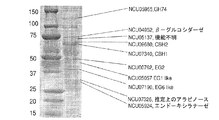
本明細書に使用されるとき、セロデキストリンは、種々の長さのグルコース重合体を指し、当該セロデキストリンとしては、セロビオース(2つのグルコースの単量体)、セロトリオース(3つのグルコースの単量体)、セロテトラオース(4つのグルコースの単量体)、セロペンタオース(5つのグルコースの単量体)およびセロヘキサオース(6つのグルコースの単量体)が挙げられるが、これらに限定されない。
したがって、一局面は、細胞へのセロデキストリンの輸送を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および当該組換えポリヌクレオチドが発現されるような培地において細胞を培養することを包含している。当該組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしている。ここで、膜貫通型のα−ヘリックス1は配列番号1を含んでいる。上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、細胞へのセロデキストリンの促進された輸送をもたらす。
他の局面は、細胞へのセロデキストリンの輸送を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および当該組換えポリヌクレオチドが発現されるような培地において細胞を培養することを包含している。当該組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしている。ここで、膜貫通型のα−ヘリックス2は配列番号2を含んでいる。上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、細胞へのセロデキストリンの促進された輸送をもたらす。
他の局面は、細胞へのセロデキストリンの輸送を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および当該組換えポリヌクレオチドが発現されるような培地において細胞を培養することを包含している。当該組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしている。ここで、膜貫通型のα−ヘリックス2および膜貫通型のα−ヘリックス3を接続しているループは配列番号3を含んでいる。上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、細胞へのセロデキストリンの促進された輸送をもたらす。
他の局面は、細胞へのセロデキストリンの輸送を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および当該組換えポリヌクレオチドが発現されるような培地において細胞を培養することを包含している。当該組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしている。ここで、膜貫通型のα−ヘリックス5は配列番号4を含んでいる。上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、細胞へのセロデキストリンの促進された輸送をもたらす。
他の局面は、細胞へのセロデキストリンの輸送を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および当該組換えポリヌクレオチドが発現されるような培地において細胞を培養することを包含している。当該組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしている。ここで、膜貫通型のα−ヘリックス6は配列番号5を含んでいる。上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、細胞へのセロデキストリンの促進された輸送をもたらす。
他の局面は、細胞へのセロデキストリンの輸送を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および当該組換えポリヌクレオチドが発現されるような培地において細胞を培養することを包含している。当該組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしている。ここで、膜貫通型のα−ヘリックス6および膜貫通型のα−ヘリックス7の間にある配列は配列番号6を含んでいる。上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、細胞へのセロデキストリンの促進された輸送をもたらす。
他の局面は、細胞へのセロデキストリンの輸送を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および当該組換えポリヌクレオチドが発現されるような培地において細胞を培養することを包含している。当該組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしている。ここで、膜貫通型のα−ヘリックス7は配列番号7を含んでいる。上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、細胞へのセロデキストリンの促進された輸送をもたらす。
他の局面は、細胞へのセロデキストリンの輸送を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および当該組換えポリヌクレオチドが発現されるような培地において細胞を培養することを包含している。当該組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしている。ここで、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびこれらの間にある配列は配列番号8を含んでいる。上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、細胞へのセロデキストリンの促進された輸送をもたらす。
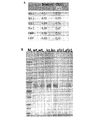
上述の局面のいずれかと組み合わせられ得る一部の実施形態において、上記ポリペプチドは、少なくとも29%、少なくとも30%、少なくとも35%、少なくとも40%、少なくとも45%、少なくとも50%、少なくとも55%、少なくとも60%、少なくとも65%、少なくとも70%、少なくとも75%、少なくとも80%、少なくとも85%、少なくとも90%、少なくとも95%、少なくとも99%または少なくとも100%の、NCU00801またはNCU08114に対するアミノ酸同一性を有している。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記宿主細胞は、Neurospora crassa由来のβ−グルコシダーゼの触媒作用ドメインを少なくともコードしている第2の組換えポリヌクレオチドを含んでいる。Neurospora crassa由来のβ−グルコシダーゼの触媒作用ドメインを少なくともコードしている第2の組換えポリヌクレオチドを含んでいる細胞を有している上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記β−グルコシダーゼはNCU00130によってコードされている。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記宿主細胞は、ペントース利用に関与する1つ以上の酵素をコードしている1つ以上の組換えポリヌクレオチドをさらに含んでいる。ペントース利用に関与する1つ以上の酵素をコードしている1つ以上の組換えポリヌクレオチドをさらに含んでいる宿主細胞を有している上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、1つ以上の上記酵素は、L−アラビノースイソメラーゼ、L−リブロキナーゼ、L−リブロース−5−P 4 エピメラーゼ、キシロースイソメラーゼ、キシルロキナーゼ、アルドースリダクターゼ、L−アラビニトール 4−デヒドロゲナーゼ、L−キシルロースリダクターゼ、およびキシトールデヒドロゲナーゼからなる群のうちの1つ以上から選択される。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記宿主細胞は、ペントーストランスポータをコードしている第3の組換えポリヌクレオチドをさらに含んでいる。、ペントーストランスポータをコードしている第3の組換えポリヌクレオチドをさらに含んでいる宿主細胞を有している上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記ペントーストランスポータは、NCU00821、NCU04963、NCU06138、STL12/XUT6、SUT2、SUT3、XUT1、およびXUT3からなる群から選択される。
他の局面は、細胞の成長を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および当該組換えポリヌクレオチドが発現されるような培地において細胞を培養することを包含している。当該組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしている。ここで、膜貫通型のα−ヘリックス1は配列番号1を含んでいる。上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。
他の局面は、細胞の成長を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および当該組換えポリヌクレオチドが発現されるような培地において細胞を培養することを包含している。当該組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしている。ここで、膜貫通型のα−ヘリックス2は配列番号2を含んでいる。上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。
他の局面は、細胞の成長を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および当該組換えポリヌクレオチドが発現されるような培地において細胞を培養することを包含している。当該組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしている。ここで、膜貫通型のα−ヘリックス2および膜貫通型のα−ヘリックス3を接続しているループは配列番号3を含んでいる。上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。
他の局面は、細胞の成長を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および当該組換えポリヌクレオチドが発現されるような培地において細胞を培養することを包含している。当該組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしている。ここで、膜貫通型のα−ヘリックス5は配列番号4を含んでいる。上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。
他の局面は、細胞の成長を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および当該組換えポリヌクレオチドが発現されるような培地において細胞を培養することを包含している。当該組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしている。ここで、膜貫通型のα−ヘリックス66は配列番号5を含んでいる。上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。
他の局面は、細胞の成長を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および当該組換えポリヌクレオチドが発現されるような培地において細胞を培養することを包含している。当該組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしている。ここで、膜貫通型のα−ヘリックス6および膜貫通型のα−ヘリックス7の間にある配列は配列番号6を含んでいる。上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。
他の局面は、細胞の成長を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および当該組換えポリヌクレオチドが発現されるような培地において細胞を培養することを包含している。当該組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしている。ここで、膜貫通型のα−ヘリックス7は配列番号7を含んでいる。上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。
他の局面は、細胞の成長を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および当該組換えポリヌクレオチドが発現されるような培地において細胞を培養することを包含している。当該組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしている。ここで、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびそれらの間にある配列は配列番号を含んでいる。上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。
上述の局面のいずれかと組み合わせられ得る一部の実施形態において、上記ポリペプチドは、少なくとも29%、少なくとも30%、少なくとも35%、少なくとも40%、少なくとも45%、少なくとも50%、少なくとも55%、少なくとも60%、少なくとも65%、少なくとも70%、少なくとも75%、少なくとも80%、少なくとも85%、少なくとも90%、少なくとも95%、少なくとも99%または少なくとも100%の、NCU00801またはNCU08114に対するアミノ酸同一性を有している。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記宿主細胞は、Neurospora crassa由来のβ−グルコシダーゼの触媒作用ドメインを少なくともコードしている外因性または第2の組換えポリヌクレオチドを含んでいる。Neurospora crassa由来のβ−グルコシダーゼの触媒作用ドメインを少なくともコードしている外因性または第2の組換えポリヌクレオチドを含んでいる細胞を有している上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記β−グルコシダーゼはNCU00130によってコードされている。
他の局面は、セルロース由来の糖およびヘミセルロース由来の糖を共発酵させる方法を包含している。当該方法は、第1の組換えポリヌクレオチドおよび第2の組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、ならびにセルロース由来の糖およびヘミセルロース由来の糖を含んでいる培地において上記宿主細胞を培養することを包含している。上記第1の組換えポリヌクレオチドはセロデキストリントランスポータをコードしており、第2の組換えポリヌクレオチドはβ−グルコシダーゼの触媒作用ドメインをコードしている。2つの上記組換えポリヌクレオチドの発現は、セルロース由来の糖およびヘミセルロース由来の糖の共発酵を可能にする。一部の実施形態において、上記第1の組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、ここで、膜貫通型のα−ヘリックス1は配列番号1を含んでいる。一部の実施形態において、上記第1の組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、ここで、膜貫通型のα−ヘリックス2は配列番号2を含んでいる。一部の実施形態において、上記第1の組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、ここで、膜貫通型のα−ヘリックス2および膜貫通型のα−ヘリックス3を接続しているループは配列番号3を含んでいる。一部の実施形態において、上記第1の組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、ここで、膜貫通型のα−ヘリックス5は配列番号4を含んでいる。一部の実施形態において、上記第1の組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、ここで、膜貫通型のα−ヘリックス6は配列番号5を含んでいる。一部の実施形態において、上記第1の組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、ここで、膜貫通型のα−ヘリックス6および膜貫通型のα−ヘリックス7は配列番号6を含んでいる。一部の実施形態において、上記第1の組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、ここで、膜貫通型のα−ヘリックス7は配列番号7を含んでいる。一部の実施形態において、上記第1の組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、ここで、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびこれらの間にある配列は配列番号8を含んでいる。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記ポリペプチドは、少なくとも29%、少なくとも30%、少なくとも35%、少なくとも40%、少なくとも45%、少なくとも50%、少なくとも55%、少なくとも60%、少なくとも65%、少なくとも70%、少なくとも75%、少なくとも80%、少なくとも85%、少なくとも90%、少なくとも95%、少なくとも99%または少なくとも100%の、NCU00801またはNCU08114に対するアミノ酸同一性を有している。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記β−グルコシダーゼはNeurospora crassに由来する。Neurospora crassに由来するβ−グルコシダーゼの触媒作用ドメインをコードしている第2の組換えポリヌクレオチドを含んでいる細胞を有している上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記β−グルコシダーゼはNCU00130によってコードされている。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記宿主細胞は、ペントース利用に関与する1つ以上の酵素をコードしている1つ以上のポリヌクレオチドをさらに含んでいる。ペントース利用に関与する1つ以上の酵素をコードしている1つ以上のポリヌクレオチドをさらに含んでいる宿主細胞を有している上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、1つ以上の上記酵素は、L−アラビノースイソメラーゼ、L−リブロキナーゼ、L−リブロース−5−P 4 エピメラーゼ、キシロースイソメラーゼ、キシルロキナーゼ、アルドースリダクターゼ、L−アラビニトール 4−デヒドロゲナーゼ、L−キシルロースリダクターゼ、およびキシトールデヒドロゲナーゼからなる群のうちの1つ以上から選択される。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記宿主細胞は、ペントーストランスポータをコードしている第3の組換えポリヌクレオチドをさらに含んでいる。ペントーストランスポータをコードしている第3の組換えポリヌクレオチドをさらに含んでいる宿主細胞を有している上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記ペントーストランスポータは、NCU00821、NCU04963、NCU06138、STL12/XUT6、SUT2、SUT3、XUT1、およびXUT3からなる群から選択される。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、セルロース由来の上記糖は、セロビオース、セロトリオースおよびセロテトラオースからなる群から選択され、ヘミセルロース由来の糖はキシロースである。
他の局面は、宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および培地において上記宿主細胞を培養することを包含している。上記組換えポリヌクレオチドは、炭化水素または炭化水素の誘導体の合成のために上記宿主細胞にセロデキストリンを輸送するポリペプチドをコードしている。当該ポリペプチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでおり、膜貫通型のα−ヘリックス1は配列番号1を含んでいる。上記培地は、上記宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、セロデキストリンまたはセロデキストリン源を含んでいる。上記宿主細胞へのセロデキストリンの輸送は、上記組換えポリヌクレオチドの発現によって促進される。
他の局面は、宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および培地において上記宿主細胞を培養することを包含している。上記組換えポリヌクレオチドは、炭化水素または炭化水素の誘導体の合成のために上記宿主細胞にセロデキストリンを輸送するポリペプチドをコードしている。当該ポリペプチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでおり、膜貫通型のα−ヘリックス2は配列番号2を含んでいる。上記培地は、上記宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、セロデキストリンまたはセロデキストリン源を含んでいる。上記宿主細胞へのセロデキストリンの輸送は、上記組換えポリヌクレオチドの発現によって促進される。
他の局面は、宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および培地において上記宿主細胞を培養することを包含している。上記組換えポリヌクレオチドは、炭化水素または炭化水素の誘導体の合成のために上記宿主細胞にセロデキストリンを輸送するポリペプチドをコードしている。当該ポリペプチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでおり、膜貫通型のα−ヘリックス2および膜貫通型のα−ヘリックス3を接続しているループは配列番号3を含んでいる。上記培地は、上記宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、セロデキストリンまたはセロデキストリン源を含んでいる。上記宿主細胞へのセロデキストリンの輸送は、上記組換えポリヌクレオチドの発現によって促進される。
他の局面は、宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および培地において上記宿主細胞を培養することを包含している。上記組換えポリヌクレオチドは、炭化水素または炭化水素の誘導体の合成のために上記宿主細胞にセロデキストリンを輸送するポリペプチドをコードしている。当該ポリペプチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでおり、膜貫通型のα−ヘリックス5は配列番号4を含んでいる。上記培地は、上記宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、セロデキストリンまたはセロデキストリン源を含んでいる。上記宿主細胞へのセロデキストリンの輸送は、上記組換えポリヌクレオチドの発現によって促進される。
他の局面は、宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および培地において上記宿主細胞を培養することを包含している。上記組換えポリヌクレオチドは、炭化水素または炭化水素の誘導体の合成のために上記宿主細胞にセロデキストリンを輸送するポリペプチドをコードしている。当該ポリペプチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでおり、膜貫通型のα−ヘリックス6は配列番号5を含んでいる。上記培地は、上記宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、セロデキストリンまたはセロデキストリン源を含んでいる。上記宿主細胞へのセロデキストリンの輸送は、上記組換えポリヌクレオチドの発現によって促進される。
他の局面は、宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および培地において上記宿主細胞を培養することを包含している。上記組換えポリヌクレオチドは、炭化水素または炭化水素の誘導体の合成のために上記宿主細胞にセロデキストリンを輸送するポリペプチドをコードしている。当該ポリペプチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでおり、膜貫通型のα−ヘリックス6および膜貫通型のα−ヘリックス7の間にある配列は配列番号6を含んでいる。上記培地は、上記宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、セロデキストリンまたはセロデキストリン源を含んでいる。上記宿主細胞へのセロデキストリンの輸送は、上記組換えポリヌクレオチドの発現によって促進される。
他の局面は、宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および培地において上記宿主細胞を培養することを包含している。上記組換えポリヌクレオチドは、炭化水素または炭化水素の誘導体の合成のために上記宿主細胞にセロデキストリンを輸送するポリペプチドをコードしている。当該ポリペプチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでおり、膜貫通型のα−ヘリックス7は配列番号7を含んでいる。上記培地は、上記宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、セロデキストリンまたはセロデキストリン源を含んでいる。上記宿主細胞へのセロデキストリンの輸送は、上記組換えポリヌクレオチドの発現によって促進される。
他の局面は、宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、および培地において上記宿主細胞を培養することを包含している。上記組換えポリヌクレオチドは、炭化水素または炭化水素の誘導体の合成のために上記宿主細胞にセロデキストリンを輸送するポリペプチドをコードしている。当該ポリペプチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでおり、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびこれらの間にある配列は配列番号8を含んでいる。上記培地は、上記宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、セロデキストリンまたはセロデキストリン源を含んでいる。上記宿主細胞へのセロデキストリンの輸送は、上記組換えポリヌクレオチドの発現によって促進される。
上述の局面のいずれかと組み合わせられ得る一部の実施形態において、上記ポリペプチドは、少なくとも29%、少なくとも30%、少なくとも35%、少なくとも40%、少なくとも45%、少なくとも50%、少なくとも55%、少なくとも60%、少なくとも65%、少なくとも70%、少なくとも75%、少なくとも80%、少なくとも85%、少なくとも90%、少なくとも95%、少なくとも99%または少なくとも100%の、NCU00801またはNCU08114に対するアミノ酸同一性を有している。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記宿主細胞は、β−グルコシダーゼの触媒作用ドメインを少なくともコードしている第2の組換えポリヌクレオチドを含んでいる。β−グルコシダーゼの触媒作用ドメインを少なくともコードしている第2の組換えポリヌクレオチドを含んでいる宿主細胞を有している上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記β−グルコシダーゼはNeurospora crassaに由来する。Neurospora crassa由来のβ−グルコシダーゼの触媒作用ドメインを少なくともコードしている第2の組換えポリヌクレオチドを含んでいる細胞を有している上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記β−グルコシダーゼはNCU00130によってコードされている。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、セロデキストリン源はセルロースを含んでいる。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記炭化水素または炭化水素の誘導体は燃料として使用され得る。燃料として使用され得る炭化水素または炭化水素の誘導体を有している上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記炭化水素または炭化水素の誘導体はエタノールを含んでいる。燃料として使用され得る炭化水素または炭化水素の誘導体を有している上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記炭化水素または炭化水素の誘導体はブタノールを含んでいる。
上述の局面のいずれかと組み合わせられ得る一部の実施形態において、上記培地は、改変された生物から得られた、セルラーゼ含有の酵素混合物を含んでおり、セルラーゼ含有の当該酵素混合物は、改変されていない生物から得られた、セルラーゼ含有の酵素混合物と比べて、低いセルラーゼ活性を有している。上述の局面のいずれかと組み合わせられ得る一部の実施形態において、上記宿主細胞は、Saccharomyces cerevisiae、Saccharomyces monacensis、Saccharomyces bayanus、Saccharomyces pastorianus、Saccharomyces carlsbergensis、Saccharomyces pombe、Kluyveromyces sp.、Kluyveromyces marxiamus、Kluyveromyces lactis、Kluyveromyces fragilis、Pichia stipitis、Sporotrichum thermophile、Candida shehatae、Candida tropicalis、Neurospora crassa、Zymomonas mobilis、Clostridium sp.、Clostridium phytofermentans、Clostridium thermocellum、Clostridium beijerinckii、Clostridium acetobutylicum、Moorella thermoacetica、Escherichia coli、Klebsiella oxytoca、Thermoanaerobacterium saccharolyticum、およびBacillus subtilisからなる群から選択される。上述の局面のいずれかと組み合わせられ得る一部の実施形態において、セロデキストリンは、セロビオース、セロトリオースおよびセロテトラオースからなる群から選択される。
他の局面は、ポリペプチドをコードしている組換えポリヌクレオチドを含んでいる細胞を包含している。当該ポリペプチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12、細胞内のN末端、細胞内のC末端、ならびに膜貫通型のα−ヘリックス1における配列番号1;膜貫通型のα−ヘリックス2における配列番号2;膜貫通型のα−ヘリックス2および膜貫通型のα−ヘリックス3を接続しているループにおける配列番号3;膜貫通型のα−ヘリックス5における配列番号4;膜貫通型のα−ヘリックス6における配列番号5;膜貫通型のα−ヘリックス6および膜貫通型のα−ヘリックス7の間にある配列における配列番号6;膜貫通型のα−ヘリックス7における配列番号7;ならびに膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびこれらの間にある配列における配列番号8からなる群から選択される配列を有している。一部の実施形態において、上記ポリペプチドは、少なくとも29%、少なくとも30%、少なくとも35%、少なくとも40%、少なくとも45%、少なくとも50%、少なくとも55%、少なくとも60%、少なくとも65%、少なくとも70%、少なくとも75%、少なくとも80%、少なくとも85%、少なくとも90%、少なくとも95%、少なくとも99%または少なくとも100%の、NCU00801またはNCU08114に対するアミノ酸同一性を有している。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記宿主細胞は、β−グルコシダーゼの触媒作用ドメインを少なくともコードしている第2の組換えポリヌクレオチドを含んでいる。β−グルコシダーゼの触媒作用ドメインを少なくともコードしている第2の組換えポリヌクレオチドを含んでいる宿主細胞を有している上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記β−グルコシダーゼはNeurospora crassaに由来する。Neurospora crassa由来のβ−グルコシダーゼの触媒作用ドメインを少なくともコードしている第2の組換えポリヌクレオチドを含んでいる細胞を有している上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記β−グルコシダーゼはNCU00130によってコードされている。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記宿主細胞は、ペントース利用に関与する1つ以上の酵素をコードしている1つ以上の組換えポリヌクレオチドをさらに含んでいる。ペントース利用に関与する1つ以上の酵素をコードしている1つ以上の組換えポリヌクレオチドをさらに含んでいる宿主細胞を有しているいくつかの実施形態において、1つ以上の上記酵素は、L−アラビノースイソメラーゼ、L−リブロキナーゼ、L−リブロース−5−P 4 エピメラーゼ、キシロースイソメラーゼ、キシルロキナーゼ、アルドースリダクターゼ、L−アラビニトール 4−デヒドロゲナーゼ、L−キシルロースリダクターゼ、およびキシトールデヒドロゲナーゼからなる群のうちの1つ以上から選択される。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記宿主細胞は、ペントーストランスポータをコードしている第3の組換えポリヌクレオチドをさらに含んでいる。ペントーストランスポータをコードしている第3の組換えポリヌクレオチドをさらに含んでいる宿主細胞を有している上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記ペントーストランスポータは、NCU00821、NCU04963、NCU06138、STL12/XUT6、SUT2、SUT3、XUT1、およびXUT3からなる群から選択される。
上述の局面のいずれかと組み合わせられ得る一部の実施形態において、上記宿主細胞は、1つ以上の上記組換えポリヌクレオチドと作動可能に連結されている1つ以上の誘導性のプロモータをさらに含んでいる。
他の局面は、NCU00821およびSTL12/XUT6からなる群から選択されるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を包含しており、当該ポリペプチドは当該宿主細胞にキシロースを輸送すする。
他の局面は、XUT1であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を包含しており、当該ポリペプチドはアラビノースを当該宿主細胞に輸送する。
他の局面は、NCU06138であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を包含しており、当該ポリペプチドはアラビノースおよびグルコースを当該宿主細胞に輸送する。
他の局面は、SUT2、SUT3およびXUT3からなる群から選択されるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を包含しており、当該ポリペプチドはキシロースおよびグルコースを当該宿主細胞に輸送する。
他の局面は、NCU04963であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を包含しており、当該ポリペプチドは、キシロース、アラビノースおよびグルコースを当該宿主細胞に輸送する。
ペントーストランスポータをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を有している上述の局面のいずれかと組み合わせられ得る一部の実施形態において、上記宿主細胞は、ペントース利用に関与する1つ以上の酵素をコードしている1つ以上の組換えポリヌクレオチドをさらに含んでいる。ペントース利用に関与する1つ以上の酵素をコードしている1つ以上の組換えポリヌクレオチドをさらに含んでいる宿主細胞を有している上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、1つ以上の上記酵素は、L−アラビノースイソメラーゼ、L−リブロキナーゼ、L−リブロース−5−P 4 エピメラーゼ、キシロースイソメラーゼ、キシルロキナーゼ、アルドースリダクターゼ、L−アラビニトール 4−デヒドロゲナーゼ、L−キシルロースリダクターゼ、およびキシトールデヒドロゲナーゼからなる群のうちの1つ以上から選択される。
他の局面は、宿主細胞へのキシロースの輸送を促進させる方法を包含している。当該方法は、NCU00821およびSTL12/XUT6からなる群から選択されるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる細胞を準備すること、ならびに上記組換えポリヌクレオチドが発現されるように上記宿主細胞を培養することを包含している。上記組換えポリヌクレオチドの発現は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記宿主細胞へのキシロースの促進された輸送をもたらす。
他の局面は、宿主細胞へのキシロースの輸送を促進させる方法を包含している。当該方法は、NCU00821およびSTL12/XUT6からなる群から選択されるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる細胞を準備すること、ならびに上記組換えポリヌクレオチドが発現されるように上記宿主細胞を培養することを包含している。上記組換えポリヌクレオチドの発現は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記宿主細胞へのキシロースの促進された輸送をもたらす。
他の局面は、宿主細胞へのアラビノースまたはグルコースの輸送を促進させる方法を包含している。当該方法は、NCU06138であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる細胞を準備すること、ならびに上記組換えポリヌクレオチドが発現されるように上記宿主細胞を培養することを包含している。上記組換えポリヌクレオチドの発現は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記宿主細胞へのアラビノースまたはグルコースの促進された輸送をもたらす。
他の局面は、宿主細胞へのキシロースまたはグルコースの輸送を促進させる方法を包含している。当該方法は、SUT2、SUT3およびXUT3からなる群から選択されるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる細胞を準備すること、ならびに上記組換えポリヌクレオチドが発現されるように上記宿主細胞を培養することを包含している。上記組換えポリヌクレオチドの発現は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記宿主細胞へのキシロースまたはグルコースの促進された輸送をもたらす。
他の局面は、宿主細胞へのキシロース、アラビノースまたはグルコースの輸送を促進させる方法を包含している。当該方法は、NCU04963であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる細胞を準備すること、ならびに上記組換えポリヌクレオチドが発現されるように上記宿主細胞を培養することを包含している。上記組換えポリヌクレオチドの発現は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記宿主細胞へのキシロース、アラビノースまたはグルコースの促進された輸送をもたらす。
細胞へのキシロース、アラビノースまたはグルコースの輸送を促進させる上述の局面のいずれかと組み合わせられ得る一部の実施形態において、上記方法は、ペントース輸送に関与する1つ以上の酵素をコードしている1つ以上の組換えポリヌクレオチドをさらに含んでいる。ペントース輸送に関与する1つ以上の酵素をコードしている1つ以上の組換えポリヌクレオチドをさらに含んでいる方法を有している上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、1つ以上の上記酵素は、L−アラビノースイソメラーゼ、L−リブロキナーゼ、L−リブロース−5−P 4 エピメラーゼ、キシロースイソメラーゼ、キシルロキナーゼ、アルドースリダクターゼ、L−アラビニトール 4−デヒドロゲナーゼ、L−キシルロースリダクターゼ、およびキシトールデヒドロゲナーゼからなる群のうちの1つ以上から選択される。
他の局面は細胞の成長を促進させる方法を包含している。当該方法は、キシロースを輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる細胞を準備すること、およびキシロースを含んでいる培地において上記宿主細胞を培養することを包含している。上記ポリペプチドは、NCU00821およびSTL12/XUT6からなる群から選択される。上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。
他の局面は細胞の成長を促進させる方法を包含している。当該方法は、アラビノースを輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる細胞を準備すること、およびアラビノースを含んでいる培地において上記宿主細胞を培養することを包含している。上記ポリペプチドはXUT1である。上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。
他の局面は細胞の成長を促進させる方法を包含している。当該方法は、アラビノースおよびグルコースを輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる細胞を準備すること、ならびにアラビノースおよびグルコースを含んでいる培地において上記宿主細胞を培養することを包含している。上記ポリペプチドは、NCU06138である。上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。
他の局面は細胞の成長を促進させる方法を包含している。当該方法は、キシロースおよびグルコースを輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる細胞を準備すること、ならびにキシロースおよびグルコースを含んでいる培地において上記宿主細胞を培養することを包含している。上記ポリペプチドは、SUT2、SUT3およびSTL12/XUT1からなる群から選択される。上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。
他の局面は細胞の成長を促進させる方法を包含している。当該方法は、キシロース、アラビノースおよびグルコースを輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる細胞を準備すること、ならびにキシロース、アラビノースおよびグルコースを含んでいる培地において上記宿主細胞を培養することを包含している。上記ポリペプチドは、NCU04963である。上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。
キシロース、アラビノースおよび/またはグルコースを輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を培養することによって細胞の成長を促進させる上述の局面のいずれかと組み合わせられ得る一部の実施形態において、上記宿主細胞は、ペントース利用に関与する1つ以上の酵素をコードしている外因性のポリヌクレオチドまたは組換えポリヌクレオチドの1つ以上をさらに含んでいる。ペントース利用に関与する1つ以上の酵素をコードしている外因性のポリヌクレオチドまたは組換えポリヌクレオチドの1つ以上をさらに含んでいる宿主細胞を有している上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、L−アラビノースイソメラーゼ、L−リブロキナーゼ、L−リブロース−5−P 4 エピメラーゼ、キシロースイソメラーゼ、キシルロキナーゼ、アルドースリダクターゼ、L−アラビニトール 4−デヒドロゲナーゼ、L−キシルロースリダクターゼ、およびキシトールデヒドロゲナーゼからなる群のうちの1つ以上から選択される。
他の局面は宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法を包含している。当該方法は、NCU00821およびSTL12/XUT6からなる群から選択されるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、ならびに当該宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、キシロースまたはキシロース源を含んでいる培地において上記宿主細胞を培養することを包含している。上記ポリペプチドは、上記宿主細胞による炭化水素または炭化水素の誘導体の合成のために当該宿主細胞にキシロースを輸送する。上記宿主細胞へのキシロースの輸送は上記組換えポリヌクレオチドの発現によって促進される。
他の局面は宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法を包含している。当該方法は、XUT1であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、ならびに当該宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、アラビノースまたはアラビノース源を含んでいる培地において上記宿主細胞を培養することを包含している。上記ポリペプチドは、上記宿主細胞による炭化水素または炭化水素の誘導体の合成のために当該宿主細胞にアラビノースを輸送する。上記宿主細胞へのアラビノースの輸送は上記組換えポリヌクレオチドの発現によって促進される。
他の局面は宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法を包含している。当該方法は、NCU06138であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、ならびに当該宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、アラビノースもしくはグルコースの源、アラビノースまたはグルコースを含んでいる培地において上記宿主細胞を培養することを包含している。上記ポリペプチドは、上記宿主細胞による炭化水素または炭化水素の誘導体の合成のために当該宿主細胞にアラビノースまたはグルコースを輸送する。上記宿主細胞へのアラビノースまたはグルコースの輸送は上記組換えポリヌクレオチドの発現によって促進される。
他の局面は宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法を包含している。当該方法は、SUT2、SUT3およびXUT3からなる群から選択されるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、ならびに当該宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、キシロースもしくはグルコース源、キシロースまたはグルコースを含んでいる培地において上記宿主細胞を培養することを包含している。上記ポリペプチドは、上記宿主細胞による炭化水素または炭化水素の誘導体の合成のために当該宿主細胞にキシロースを輸送する。上記宿主細胞へのキシロースの輸送は上記組換えポリヌクレオチドの発現によって促進される。
他の局面は宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法を包含している。当該方法は、NCU00821およびSTL12/XUT6からなる群から選択されるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、ならびに当該宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、キシロース、アラビノースもしくはグルコース源、キシロース、アラビノースまたはグルコースを含んでいる培地において上記宿主細胞を培養することを包含している。上記ポリペプチドは、上記宿主細胞による炭化水素または炭化水素の誘導体の合成のために当該宿主細胞にキシロース、アラビノースまたはグルコースを輸送する。上記宿主細胞へのキシロース、アラビノースまたはグルコースの輸送は、上記組換えポリヌクレオチドの発現によって促進される。
グルコースを輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を培養することによって炭化水素または炭化水素の誘導体の合成を促進させる上述の局面のいずれかと組み合わせられ得る一部の実施形態において、上記グルコース源はセルロースを含んでいる。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、キシロースまたはアラビノースの上記源はヘミセルロースを含んでいる。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記炭化水素または炭化水素の誘導体は燃料として使用され得る。燃料として使用され得る上記炭化水素または炭化水素の誘導体を有している上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記炭化水素または炭化水素の誘導体はエタノール含んでいる。燃料として使用され得る上記炭化水素または炭化水素の誘導体を有している上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記炭化水素または炭化水素の誘導体はブタノールを含んでいる。
上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記宿主細胞は、Saccharomyces sp.、Saccharomyces cerevisiae、Saccharomyces monacensis、Saccharomyces bayanus、Saccharomyces pastorianus、Saccharomyces carlsbergensis、Saccharomyces pombe、Kluyveromyces sp.、Kluyveromyces marxiamus、Kluyveromyces lactis、Kluyveromyces fragilis、Pichia stipitis、Sporotrichum thermophile、Candida shehatae、Candida tropicalis、Neurospora crassa、Zymomonas mobilis、Clostridium sp.、Clostridium phytofermentans、Clostridium thermocellum、Clostridium beijerinckii、Clostridium acetobutylicum、Moorella thermoacetica、Escherichia coli、Klebsiella oxytoca、Thermoanaerobacterium saccharolyticum、およびBacillus subtilisからなる群から選択される。
他の局面は細胞の成長を促進させる方法を包含している。当該方法は、NCU07705であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる細胞を準備すること、ならびにセルロースを含んでいる培地において当該宿主細胞を培養することを包含している。上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。一部の実施形態において、上記宿主細胞は、Saccharomyces sp.、Saccharomyces cerevisiae、Saccharomyces monacensis、Saccharomyces bayanus、Saccharomyces pastorianus、Saccharomyces carlsbergensis、Saccharomyces pombe、Kluyveromyces sp.、Kluyveromyces marxiamus、Kluyveromyces lactis、Kluyveromyces fragilis、Pichia stipitis、Sporotrichum thermophile、Candida shehatae、Candida tropicalis、Neurospora crassa、Zymomonas mobilis、Clostridium sp.、Clostridium phytofermentans、Clostridium thermocellum、Clostridium beijerinckii、Clostridium acetobutylicum、Moorella thermoacetica、Escherichia coli、Klebsiella oxytoca、Thermoanaerobacterium saccharolyticum、およびBacillus subtilisからなる群から選択される。一部の実施形態において、上記宿主細胞は、上記組換えポリヌクレオチドと作動可能に連結されている誘導性のプロモータをさらに含んでいる。一部の実施形態において、セルラーゼの発現は上記組換えポリヌクレオチドの発現によって宿主細胞において促進される。
他の局面は、バイオマス重合体に基づく細胞の成長を促進させる方法を包含している。当該方法は、NCU05137であるポリペプチドをコードしている外因性のポリヌクレオチドを含んでいる細胞を準備すること、当該外因性のポリヌクレオチドの発現を抑制すること、ならびにバイオマス重合体を含んでいる培地において当該宿主細胞を培養することを包含している。上記宿主細胞は、上記外因性のポリヌクレオチドの発現が抑制されていない細胞と比べて、上記培地においてより早く成長する。一部の実施形態において、上記宿主細胞は、Saccharomyces sp.、Saccharomyces cerevisiae、Saccharomyces monacensis、Saccharomyces bayanus、Saccharomyces pastorianus、Saccharomyces carlsbergensis、Saccharomyces pombe、Kluyveromyces sp.、Kluyveromyces marxiamus、Kluyveromyces lactis、Kluyveromyces fragilis、Pichia stipitis、Sporotrichum thermophile、Candida shehatae、Candida tropicalis、Neurospora crassa、Zymomonas mobilis、Clostridium sp.、Clostridium phytofermentans、Clostridium thermocellum、Clostridium beijerinckii、Clostridium acetobutylicum、Moorella thermoacetica、Escherichia coli、Klebsiella oxytoca、Thermoanaerobacterium saccharolyticum、およびBacillus subtilisからなる群から選択される。一部の実施形態において、上記宿主細胞のセルラーゼ活性は、上記外因性のポリヌクレオチドの発現を抑制することによって向上されている。一部の実施形態において、上記宿主細胞のヘミセルラーゼ活性は、上記外因性のポリヌクレオチドの発現を抑制することによって向上されている。一部の実施形態において、上記組換えポリヌクレオチドの発現を抑制することは、上記組換えポリヌクレオチドを含んでいる遺伝子を変異させること、または当該遺伝子を欠失させることを包含している。一部の実施形態において、上記バイオマス重合体はセルロースである。一部の実施形態において、上記バイオマス重合体はヘミセルロースである。
他の局面は細胞の成長を促進させる方法を包含している。当該方法は、NCU01517、NCU09133およびNCU10040からなる群から選択されるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる細胞を準備すること、ならびにセルロースを含んでいる培地において当該宿主細胞を培養することを包含している。上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。一部の実施形態において、上記宿主細胞は、Saccharomyces sp.、Saccharomyces cerevisiae、Saccharomyces monacensis、Saccharomyces bayanus、Saccharomyces pastorianus、Saccharomyces carlsbergensis、Saccharomyces pombe、Kluyveromyces sp.、Kluyveromyces marxiamus、Kluyveromyces lactis、Kluyveromyces fragilis、Pichia stipitis、Sporotrichum thermophile、Candida shehatae、Candida tropicalis、Neurospora crassa、Zymomonas mobilis、Clostridium sp.、Clostridium phytofermentans、Clostridium thermocellum、Clostridium beijerinckii、Clostridium acetobutylicum、Moorella thermoacetica、Escherichia coli、Klebsiella oxytoca、Thermoanaerobacterium saccharolyticum、およびBacillus subtilisからなる群から選択される。一部の実施形態において、上記宿主細胞は、上記組換えポリヌクレオチドと作動可能に連結されている誘導性のプロモータをさらに含んでいる。一部の実施形態において、ヘミセルラーゼ活性は、上記組換えポリヌクレオチドの発現によって向上されている。
他の局面はセルロースを分解させる方法を包含している。当該方法は、セルロースを含んでいる組成物を準備すること、ならびに当該組成物を、改変された生物から得られた、セルラーゼ含有の酵素混合物と接触させることを包含している。セルラーゼ含有の上記酵素混合物は、改変されていない生物から得られた、セルラーゼ含有の酵素混合物と比べて、低下したβ−グルコシダーゼ活性を有しており、上記セルロースは、セルラーゼ含有の上記酵素混合物によって分解される。一部の実施形態において、上記生物はβ−グルコシダーゼをコードしている遺伝子の変異によって改変されている。一部の実施形態において、上記生物はβ−グルコシダーゼの発現を低下させることによって改変されている。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記生物は真菌および細菌からなる群から選択される。真菌および細菌からなる群から選択される上記生物を有している上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記生物は糸状菌である。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記セルロースは植物材料に由来する。植物材料に由来するセルロースを有している上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記植物材料は、スイッチグラス、Miscanthus、もみ殻、バガス、アマ、タケ、サイザル繊維、アバカ、麦わら、葉、刈り取った芝、トウモロコシの茎および葉、トウモロコシの穂軸、穀類の蒸留粕、マメ科の植物、サトウモロコシ、サトウキビ、テンサイの果肉、材木の切り屑、おが屑、ならびにバイオマス収穫物からなる群から選択される。
他の局面は宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、ならびに当該宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるための化合物の原料を含んでいる培地において当該宿主細胞を培養することを包含している。上記組換えポリヌクレオチドは、NCU00801、NCU00988、NCU01231、NCU04963、NCU05519、NCU05853、NCU05897、NCU06138、NCU00809、NCU08114、NCU10021および表15に記載の遺伝子のうちのいずれかからなる群から選択される配列によってコードされているポリペプチドをコードしている。上記宿主細胞への上記化合物の輸送は上記組換えポリヌクレオチドの発現によって促進される。一部の実施形態において、上記宿主細胞は、Saccharomyces cerevisiae、Escherichia coli、Zymomonas mobilis、Neurospora crassa、Candida shehatae、Clostridium sp.、Clostridium phytofermentans、Clostridium thermocellum、Moorella thermocetica、Thermoanaerobacterium saccharolyticum、Klebsiella oxytoca、およびPichia stipitisからなる群から選択される。一部の実施形態において、上記宿主細胞は、上記組換えポリヌクレオチドと作動可能に連結されている誘導性のプロモータをさらに含んでいる。一部の実施形態において、上記組換えポリヌクレオチドは、NCU00801、NCU00988、NCU01231、NCU04963、NCU05519、NCU05853、NCU05897、NCU06138、NCU00809、NCU08114、NCU10021、および表15に記載のいずれかの遺伝子からなる群から選択される配列によってコードされているポリペプチドに対する少なくとも50%のアミノ酸同一性を有しているポリペプチドをコードしている。いくつかの実施形態において、上記炭化水素または炭化水素の誘導体は燃料として使用され得る。一部の実施形態において、上記培地はセルロースを含んでいる。他の実施形態において、上記培地はヘミセルロースを含んでいる。一部の実施形態において、上記化合物は糖である。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記糖はペントースである。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記糖はヘキソースである。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記糖は二糖である。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記糖はオリゴ糖である。他の実施形態において、上記化合物は植物性のフェノールである。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記植物性のフェノールはキナ酸である。上述の実施形態のいずれかと組み合わせられ得る一部の実施形態において、上記植物性のフェノールはニコチンアミドである。他の実施形態において、上記化合物はピルビン酸または乳酸である。
他の局面はバイオマス重合体に基づく細胞の成長を促進させる方法を包含している。当該方法は、組換えポリヌクレオチドを含んでいる宿主細胞を準備すること、ならびに上記バイオマス重合体を含んでいる培地において上記宿主細胞を培養することを包含している。上記組換えポリヌクレオチドは、表10に記載のNeurosporaもしくはPichia stipitisの遺伝子、Tian et al., PNAS, 2009, vol. 106, no. 52, 22157-22162(その開示事項は参照によって本明細書に援用される)における第3頁のSupplemental Data、Dataset S1に記載のNeurosporaもしくはPichia stipitisの遺伝子、表15に記載のNeurosporaもしくはPichia stipitisの遺伝子、またはNCU01517、NCU09133もしくはNCU10040によってコードされているポリペプチドをコードしている。上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、より早く成長する。一部の実施形態において、上記組換えポリヌクレオチドは、NCU00130.2、NCU00248.2、NCU00326.2、NCU00762.2、NCU00810.2、NCU00890.2、NCU03328.2、NCU03415.2、NCU03731.2、NCU03753.2、NCU04197.2、NCU04249.2、NCU04287.2、NCU04349.2、NCU04475.2、NCU04997.2、NCU05057.2、NCU05159.2、NCU05493.2、NCU05751.2、NCU05770.2、NCU05932.2、NCU06009.2、NCU06490.2、NCU07340.2、NCU07853.2、NCU07997.2、NCU08744.2、NCU08746.2、NCU08760.2、NCU09108.2、NCU09495.2、NCU09680.2、またはNCU10045.2の配列のいずれかによってコードされているポリペプチドをコードしている。一部の実施形態において、上記組換えポリヌクレオチドは、NCU07705によってコードされているポリペプチドをコードしている。一部の実施形態において、上記組換えポリヌクレオチドは、表10に記載のNeurosporaもしくはPichia stipitisの遺伝子、Tian et al., 2009における第3頁のSupplemental Data, Dataset S1に記載のNeurosporaもしくはPichia stipitisの遺伝子、または表15に記載のNeurosporaもしくはPichia stipitisの遺伝子のいずれかによってコードされているポリペプチドに対する少なくとも50%のアミノ酸同一性を有しているポリペプチドをコードしている。一部の実施形態において、上記組換えポリヌクレオチドは、NCU00130.2、NCU00248.2、NCU00326.2、NCU00762.2、NCU00810.2、NCU00890.2、NCU03328.2、NCU03415.2、NCU03731.2、NCU03753.2、NCU04197.2、NCU04249.2、NCU04287.2、NCU04349.2、NCU04475.2、NCU04997.2、NCU05057.2、NCU05159.2、NCU05493.2、NCU05751.2、NCU05770.2、NCU05932.2、NCU06009.2、NCU06490.2、NCU07340.2、NCU07853.2、NCU07997.2、NCU08744.2、NCU08746.2、NCU08760.2、NCU09108.2、NCU09495.2、NCU09680.2、またはNCU10045.2の配列のいずれかによってコードされているポリペプチドに対する少なくとも50%のアミノ酸同一性を有しているポリペプチドをコードしている。一部の実施形態において、上記組換えポリヌクレオチドは、NCU07705によってコードされているポリペプチドに対する少なくとも50%のアミノ酸同一性を有しているポリペプチドをコードしている。一部の実施形態において、上記バイオマス重合体はセルロースである。一部の実施形態において、上記バイオマス重合体はヘミセルロースである。一部の実施形態において、上記宿主細胞は、Saccharomyces cerevisiae、Escherichia coli、Zymomonas mobilis、Neurospora crassa、Candida shehatae、Clostridium sp.、Clostridium phytofermentans、Clostridium thermocellum、Moorella thermocetica、Thermoanaerobacterium saccharolyticum、Klebsiella oxytoca、およびPichia stipitisからなる群から選択される。一部の実施形態において、上記宿主細胞は、上記組換えポリヌクレオチドと作動可能に連結されている誘導性のプロモータをさらに含んでいる。一部の実施形態において、セルラーゼの発現は上記組換えポリヌクレオチドの発現によって上記宿主細胞において向上されている。他の実施形態において、ヘミセルラーゼの発現は上記組換えポリヌクレオチドの発現によって上記宿主細胞において向上されている。
他の局面はバイオマス重合体に基づく細胞の成長を促進させる方法を包含している。当該方法は、外因性のポリヌクレオチドを含んでいる宿主細胞を準備すること、ならびに上記バイオマス重合体を含んでいる培地において上記宿主細胞を培養することを包含している。上記宿主細胞は、上記外因性のポリヌクレオチドが抑制されていない細胞と比べて、上記培地においてより早く成長する。上記外因性のポリヌクレオチドは、表10に記載のNeurosporaもしくはPichia stipitisの遺伝子、Tian et al., PNAS, 2009における第3頁のSupplemental Data、Dataset S1に記載のNeurosporaもしくはPichia stipitisの遺伝子、表15に記載のNeurosporaもしくはPichia stipitisの遺伝子、または上記外因性のポリヌクレオチドの発現を抑制するNeurosporaもしくはPichia stipitisの遺伝子のうちのいずれかによってコードされているポリペプチドをコードしている。一部の実施形態において、上記外因性のポリヌクレオチドは、NCU00130.2、NCU00248.2、NCU00326.2、NCU00762.2、NCU00810.2、NCU00890.2、NCU03328.2、NCU03415.2、NCU03731.2、NCU03753.2、NCU04197.2、NCU04249.2、NCU04287.2、NCU04349.2、NCU04475.2、NCU04997.2、NCU05057.2、NCU05159.2、NCU05493.2、NCU05751.2、NCU05770.2、NCU05932.2、NCU06009.2、NCU06490.2、NCU07340.2、NCU07853.2、NCU07997.2、NCU08744.2、NCU08746.2、NCU08760.2、NCU09108.2、NCU09495.2、NCU09680.2、またはNCU10045.2の配列のいずれかによってコードされているポリペプチドをコードしている。一部の実施形態において、上記外因性のポリヌクレオチドは、NCU05137によってコードされているポリペプチドをコードしている。一部の実施形態において、上記組換えポリヌクレオチドは、表10に記載のNeurosporaもしくはPichia stipitisの遺伝子、Tian et al., PNAS, 2009における第3頁のSupplemental Data、Dataset S1に記載のNeurosporaもしくはPichia stipitisの遺伝子または表15に記載のNeurosporaもしくはPichia stipitisの遺伝子のいずれかによってコードされているポリペプチドに対する少なくとも50%のアミノ酸同一性を有しているポリペプチドをコードしている。一部の実施形態において、上記外因性のポリヌクレオチドは、NCU00130.2、NCU00248.2、NCU00326.2、NCU00762.2、NCU00810.2、NCU00890.2、NCU03328.2、NCU03415.2、NCU03731.2、NCU03753.2、NCU04197.2、NCU04249.2、NCU04287.2、NCU04349.2、NCU04475.2、NCU04997.2、NCU05057.2、NCU05159.2、NCU05493.2、NCU05751.2、NCU05770.2、NCU05932.2、NCU06009.2、NCU06490.2、NCU07340.2、NCU07853.2、NCU07997.2、NCU08744.2、NCU08746.2、NCU08760.2、NCU09108.2、NCU09495.2、NCU09680.2、またはNCU10045.2の配列のいずれかによってコードされているポリペプチドに対する少なくとも50%のアミノ酸同一性を有しているポリペプチドをコードしている。一部の実施形態において、上記組換えポリヌクレオチドは、NCU05137によってコードされているポリペプチドに対する少なくとも50%のアミノ酸同一性を有しているポリペプチドをコードしている、一部の実施形態において、上記宿主細胞は、Saccharomyces cerevisiae、Escherichia coli、Zymomonas mobilis、Neurospora crassa、Candida shehatae、Clostridium sp.、Clostridium phytofermentans、Clostridium thermocellum、Moorella thermocetica、Thermoanaerobacterium saccharolyticum、Klebsiella oxytoca、およびPichia stipitisからなる群から選択される。一部の実施形態において、上記バイオマス重合体はセルロースである。他の実施形態において、上記バイオマス重合体はヘミセルロースである。一部の実施形態において、上記宿主細胞のセルラーゼ活性は、上記外因性のポリヌクレオチドの発現を抑制することによって向上されている。他の実施形態において、上記宿主細胞のヘミセルラーゼ活性は、上記外因性のポリヌクレオチドの発現を抑制することによって向上されている。一部の実施形態において、上記外因性のポリヌクレオチドの発現を抑制することは、上記外因性のポリヌクレオチドを含んでいる遺伝子を変異させること、または当該遺伝子を欠失させることを包含している。
本開示は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞に関し、ここで、以下の1つ以上:膜貫通型のα−へリックス1は配列番号1を含んでいる、膜貫通型のα−へリックス2は配列番号2を含んでいる、膜貫通型のα−ヘリックス2および膜貫通型のα−ヘリックス3を接続しているループは配列番号3を含んでいる、膜貫通型のα−へリックス5は配列番号4を含んでいる、膜貫通型のα−へリックス6は配列番号5を含んでいる、膜貫通型のα−へリックス7は配列番号7を含んでいる、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11およびそれらの間の配列は配列番号8を含んでいる、が満たされており、上記ポリペプチドは、細胞にセロデキストリンを輸送する。本明細書にさらに記載されているのは、細胞へのセロデキストリンの輸送を促進させる方法、セルロース由来の糖およびヘミセルロース由来の糖を共発酵させる方法、ならびに宿主細胞を用いて炭化水素または炭化水素の誘導体を製造する方法である。本明細書にさらに記載されているのは、細胞にペントースを輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞、細胞へのペントースの輸送を促進させる方法、ペントース糖を含んでいる培地における細胞の成長を促進させる方法、および細胞にペントースを輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備することによって炭化水素または炭化水素の誘導体を製造する方法である。
本明細書に使用されるとき、セロデキストリンは、種々の長さのグルコース重合体を指し、セロビオース(2つのグルコースの単量体)、セロトリオース(3つのグルコースの単量体)、セロテトラオース(4つのグルコースの単量体)、セロペンタオース(5つのグルコースの単量体)およびセロヘキサオース(6つのグルコースの単量体)が挙げられるが、これらに限定されない。
本明細書に使用されるとき、糖は、単糖(例えば、グルコース、フルクトース、ガラクトース、キシロース、アラビノース)、二糖(例えば、セロビオース、スクロース、ラクトース、マルトース)、およびオリゴ糖(典型的に3〜10の単糖を含んでいる)を指す。
〔本発明のポリヌクレオチド〕
本明細書における発明は、宿主細胞および当該宿主細胞を使用する方法に関し、ここで、当該宿主細胞は、種々の糖を輸送可能なポリペプチドをコードしている組換えポリヌクレオチドを含んでいる。
本明細書における発明は、宿主細胞および当該宿主細胞を使用する方法に関し、ここで、当該宿主細胞は、種々の糖を輸送可能なポリペプチドをコードしている組換えポリヌクレオチドを含んでいる。
本明細書に使用されるとき、“ポリヌクレオチド”、“核酸配列”、“核酸の配列”、およびそれらの変化形は、ポリデオキシリボヌクレオチド(2−デオキシ−D−リボースを含んでいる)、ポリリボヌクレオチド(D−リボースを含んでいる)、プリンまたはピリミジン塩基のN−グリコシドである任意の他の種類のポリヌクレオチド、ならびにDNAおよびRNAに見出だされるような、塩基の対合および塩基の積み重なりを可能にする構成において核酸塩基を含んでいるときの非ヌクレオチドの骨格を含んでいる他の重合体を総称する。したがってこれらの用語は知られている種類の核酸配列の修飾を包含している。当該修飾は、例えば、天然に存在する1つ以上のヌクレオチドの、類似物を用いた置換;例えば、ヌクレオチド内修飾(例えば、無電荷の結合(例えば、メチルホスホン酸塩、ホスホトリエステル、ホスホラミダート、カルバミン酸塩など)を有している、負電荷の結合(例えば、ホスホロチオアート、ホスホロジチオアートなど)を有している、および正電荷の結合(例えば、ホスホラミジン酸アミノアルキル、アミノアルキルホスホトリエステル)を用いた修飾など)を有している;ペンダント部分(例えば、タンパク質(ヌクレアーゼ、トキシン、抗体、シグナルペプチド、ポリ−L−リジンなどが挙げられる))を含んでいる修飾を有している;挿入物(例えばアクリジン、ソラレンなど)を有している;およびキレート剤(例えば金属、放射性金属、ホウ素、酸化金属など)を含んでいる修飾である。本明細書に使用されるとき、ヌクレオチドおよびポリヌクレオチドの記号は、生化学命名法のIUPAC−IUB委員会によって推奨されている記号である(Biochem. 9:4022, 1970)。
本明細書に使用されるとき、“ポリペプチド”は、重合されている連続する複数のアミノ酸残基(例えば少なくとも約15の連続する重合されているアミノ酸残基、必要に応じて少なくとも約30の連続する重合されているアミノ酸残基、少なくとも約50の連続する重合されているアミノ酸残基)を含んでいるアミノ酸配列である。多くの場合に、ポリペプチドは、トランスポータ、転写因子、未知の機能の未知のタンパク質、またはそれらのドメイン、部分もしくは断片である、重合されているアミノ酸残基の配列を含んでいる。トランスポータは、イオン、小分子、または炭化水素のような巨大分子の、生物学的な膜を通過する移動に関与する。転写因子は、遺伝子発現を制御可能であり、宿主細胞における遺伝子発現を促進させ得るか、または低下させ得る。ポリペプチドは、修飾されているアミノ酸残基、コドンによってコードされていない天然に存在するアミノ酸残基、および非天然に存在するアミノ酸残基を必要に応じて含んでいる。
本明細書に使用されるとき、“タンパク質”は、天然に存在するか、または合成の、アミノ酸配列、オリゴペプチド、ペプチド、ポリペプチド、またはそれらの部分を指す。
本発明の組換えポリヌクレオチドは、表10、Tian et al., 2009における第3頁のSupplemental Data, Dataset S1、表14、15、16、29、または図76に挙げられている遺伝子のうちのいずれかによってコードされているポリペプチドをコードしている任意のポリヌクレオチドを含んでいる。好ましい実施形態において、本発明のポリヌクレオチドは、以下の配列:NCU00801、NCU00809、NCU08114、NCU00130、NCU00821、NCU04963、NCU06138、STL12/XUT6、SUT2、SUT3、XUT1、XUT3、NCU07705、NCU05137、NCU01517、NCU09133またはNCU10040のうちのいずれかによってコードされているポリペプチドをコードしている任意のポリヌクレオチドを含んでいる。
一部の実施形態において、本発明の組換えポリヌクレオチドは、少なくとも約20%、少なくとも約29%、少なくとも約30%、少なくとも約40%、少なくとも約50%、少なくとも約55%、少なくとも約60%、少なくとも約65%、少なくとも約70%、少なくとも約75%、少なくとも約80%、少なくとも約85%、少なくとも約90%、少なくとも約92%、少なくとも約94%、少なくとも約96%、少なくとも約98%、少なくとも約99%または少なくとも約100%の、表10、Tian et al., 2009における第3頁のSupplemental Data, Dataset S1、表14、15、16、29、または図76に挙げられている遺伝子の列挙されている任意の遺伝子によってコードされているポリペプチドに対するアミノ酸残基配列の同一性を有しているポリペプチドをコードしている。好ましい実施形態において、本発明のポリヌクレオチドは、少なくとも約20%、少なくとも約29%、少なくとも約30%、少なくとも約40%、少なくとも約50%、少なくとも約55%、少なくとも約60%、少なくとも約65%、少なくとも約70%、少なくとも約75%、少なくとも約80%、少なくとも約85%、少なくとも約90%、少なくとも約92%、少なくとも約94%、少なくとも約96%、少なくとも約98%、少なくとも約99%または少なくとも約100%の、NCU00801、NCU00809、NCU08114、NCU00130、NCU00821、NCU04963、NCU06138、STL12/XUT6、SUT2、SUT3、XUT1、XUT3、NCU07705、NCU05137、NCU01517、NCU09133またはNCU10040の任意の配列によってコードされているポリペプチドに対するアミノ酸残基配列の同一性を有しているポリペプチドをコードしている。
本発明のポリペプチドは、以上において挙げられている遺伝子によってコードされているポリペプチドの保存的に修飾されたバリアントをコードしているポリヌクレオチドをさらに含んでいる。本明細書に使用されるとき、“保存的に修飾された変異”は、化学的に類似するアミノ酸へのあるアミノ酸の置換を生じるポリペプチドに対する個々の置換、欠失または付加を包含している。機能的に類似のアミノ酸をもたらす保存的な置換の一覧は当業者によく知られている。そのような保存的に修飾された置換は、開示されているものの多形のバリアント、種間の相同物および対立形質であり、これらのを除外しない。以下の8つの群:(1)アラニン(A)、グリシン(G);(2)アスパラギン酸(D)、グルタミン酸(E);(3)アスパラギン(N)、グルタミン(Q);(4)アルギニン(R)、リジン(K);(5)イソロイシン(I)、ロイシン(L)、メチオニン(M)、バリン(V);(6)フェニルアラニン(F)、チロシン(Y)、トリプトファン(W)、(7)セリン(S)、スレオニン(T);および(8)システイン(C)、メチオニン(M)は、互いに保存的な置換体であるアミノ酸を含んでいる(例えばCreighton, Proteins (1984)を参照)。
本発明のポリヌクレオチドは、表10、Tian et al., 2009における第3頁のSupplemental Data, Dataset S1、表14、15、16、29、または図76に挙げられている任意の遺伝子によってコードされているポリペプチドの相同物または相同分子種をコードしているポリヌクレオチドをさらに含んでいる。本明細書に使用されるとき、“相同性”は、基準配列と第2の配列の少なくとも断片との間における配列の類似性を指す。相同物は、単一の第2の配列、配列の断片または配列のデータベースに対して基準配列を比較するための、当該分野に公知の任意の方法、好ましくはBLASTツールを用いることによって特定され得る。後述されている通り、BLASTは、同一性および類似性の百分率に基づいて配列を比較する。本明細書に使用されるとき、“相同分子種性”は、共通する祖先の遺伝子に由来する、異なる種における遺伝子を指す。
2つ以上の核酸配列またはポリペプチド配列の関係における“同一”または“同一性”百分率という用語は、同一である2つ以上の配列または部分配列を指す。後述する配列比較の演算手順のうちの1つを用いてか、もしくは手動の整列化と視覚的な検査とによって評価されるときに指定される領域の全体、または比較ウインドウの全体にわたる最大の一致性のために比較され、整列化されたときに、2つの配列は、2つの配列が、同一であるアミノ酸残基またはヌクレオチドの特定の百分率(すなわち29%の同一性、任意に30%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、99%、または100%の同一性)を有している場合に、“実質的に同一”である。必要に応じて、同一性は、少なくとも約50ヌクレオチド(または10アミノ酸)の長さである領域の全体にわたってか、またはより好ましくは100〜500ヌクレオチドの長さ、1000ヌクレオチドの長さ、または1000ヌクレオチドを超える長さ(または20アミノ酸の長さ、50アミノ酸の長さ、200アミノ酸の長さまたは200アミノ酸を超えるの長さ)の領域の全体わたってに存在している。
配列比較のために、1つの配列は、試験配列が比較される基準配列としての役割を典型的に果たす。配列比較の演算手順を用いるとき、試験配列および基準配列は、コンピュータに入力され、必要に応じて部分配列調整が指示され、配列の演算手順プログラムの変数が指示される。初期設定のプログラムのパラメータが使用され得るか、または代替的なパラメータが指示され得る。それから、配列比較の演算手順は、プログラムのパラメータに基づいて、基準配列に対する試験配列にとっての配列同一性の百分率を算出する。2つの配列を同一性について比較するとき、配列は必ずしも連続している必要はないが、任意のギャップは、全体の同一性の百分率を低下させる不利な条件をもたらす。blastnにとって、初期値の変数は、Gap開始ペナルティ=5およびGap伸長ペナルティ=2である。blastpにとって、初期値のパラメータはGap開始ペナルティ=11およびGap伸長ペナルティ=1である。
本明細書に使用されるとき、“比較ウィンドウ”は、連続する位置の数(2つの配列が最適に整列化された後に、連続する位置の同じ数の基準配列と配列が比較され得る20〜600、一般的には約50〜約200、より一般的には約100〜約150が挙げられるが、これらに限定されない)のいずれか1つの部分に対する参照を包含している。比較のための配列の整列化の方法は当該技術において公知である。比較のための配列の最適な整列化は、例えば、Smith and Waterman (1981)の局所的な同一性の演算手順、Needleman and Wunsch (1970) J Mol Biol 48 (3):443−453の同一性整列化の演算手順、Pearson and Lipman (1988) Proc Natl Acad Sci USA 85 (8) : 2444−2448の類似性の方法のための検索、これらの演算手順の機械化された実施(Wisconsin Genetics Software PackageにおけるGAP、BESTFIT、FASTAおよびFASTA(Genetics Computer Group, 575 Science Dr., Madison, WI))、または手動の整列化および視覚的な検査によって、実施され得る(例えば、Brent et al., (2003) Current Protocols in Molecular Biology, John Wiley & Sons, Inc. (Ringbou Ed)を参照)。
配列同一性および配列類似性の百分率を決定するために好適な演算手順の2つの例は、Altschul et al. (1997) Nucleic Acids Res 25 (17):3389-3402およびAltschul et al. (1990) J. Mol Biol 215 (3)-403-410にそれぞれ記載されている、BLASTおよびBLAST2.0の演算手順である。BLAST解析を実施するためのソフトウェアは、全米バイオテクノロジー情報センターを通じて公然と入手可能である。この演算手順は、データベース配列における同じ長さのワードとともに整列化されたときに正とみなされるいくつかの閾値Tに一致するか、または閾値Tを満たすクエリ配列における長さWの短いワードを識別することによって、高得点の配列対(HSPs)を特定することに第1に関する。Tは、近接するワードの得点の閾値と呼ばれている(上述のAltschul et al.,)。これらの初期の近接するワードのヒット(word hits)は、それらを含んでいるより長いHSPsを検出するための検索を開始させるのためのもととして機能する。ワードのヒットは、累積の整列化得点が上昇し得る限り、各配列に沿って両方向に伸長される。累積の得点は、ヌクレオチドの配列について、変数M(整合する残基の対にとっての報酬得点;常に0を超える)および変数N(整合しない残基のペナルティ得点;常に0未満)を用いて算出される。アミノ酸配列について、採点法のマトリクスは、累積の得点を算出するために使用される。ワードのヒットの各方向への伸長は、累積の整列化得点が最大達成値から数量Xまで低下したとき;累積の得点が1つ以上の負に得点される残基の整列化によって0以下に達するとき;または配列のいずれかの端部に達したときに、停止される。BLAST演算手順の変数W、T、およびXは、整列化の感度および速度を決定する。BLASTNプログラム(ヌクレオチド配列用)は、11のワード長(W)、10の期待値(E)、M=5、N=−4、および両方の鎖の比較を、初期設定として使用する。アミノ酸配列用のBLASTPプログラムは、3のワード長(W)、10の期待値(E)、およびBLOUSUM62採点法マトリクス(Henikoff and Henikoff, (1992) Proc Natl Acad Sci USA 89 (22):10915-10919を参照)50のアラインメント、10の期待値(E)、M=5、N=−4および両方の鎖の比較を使用する。
また、BLAST演算手順は、2つの配列の間における類似性の統計分析を実施する(例えば、Karlin and Altschul, (1993) Proc Natl Acad Sci USA 90 (12):5873-5877を参照)。BLAST演算手順によってもたらされる類似性の1つの基準は、2つのヌクレオチド配列またはアミノ酸配列の間における整合が偶然に生じる確率の指標を与え得る最小合計確率(smallest sum probability)(P(N))である。例えば、基準配列に対する試験核酸の比較における最小合計確率が、約0.2未満、より好ましくは約0.01未満、および最も好ましくは0.001である場合に、核酸は基準配列と類似すると見做される。
上述の配列同一性の百分率以外の、2つの核酸配列またはポリペプチドが実質的に同一であることの他の指標は、後述されるように、第1の核酸によってコードされているポリペプチドが、第2の核酸によってコードされているポリペプチドに対して生じた抗体と免疫学的に交差反応性であることである。したがって、例えば、2つのペプチドが保存的な置換のみによって異なっている場合、典型的に、ポリペプチドは第2のポリペプチドと実質的に同一である。2つの核酸配列が実質的に同一であることの他の指標は、後述されるされるように、2つの分子またはそれらの相補物がストリンジェントな条件の下にハイブリダイズすることである。2つの核酸配列が実質的に同一であるというさらに他の証拠は、同じプライマーが配列の増幅に用いられ得るということである。
本明細書に記載の通り、本発明のポリヌクレオチドは、メジャーファシリテータスーパーファミリー(Major Facilitator Superfamily)の糖トランスポータファミリー(NCU00988、NCU10021、NCU04963、NCU06138、NCU00801、NCU08114およびNCU05853が挙げられる)を包含している。トランスポータのメジャーファシリテータスーパーファミリー(MFS)のメンバー(トランスポータ分類番号 2.A.1)のほとんどすべては、細胞内のN末端およびC末端とともに、膜貫通型の12のα−ヘリックスからなっている(S. S. Pao, I. T. Paulsen, M. H. Saier, Jr., Microbiol Mol Biol Rev 62, 1 (Mar, 1998))。MFSトランスポータの1次配列は非常に異なっている一方で、MFSトランスポータのすべてはE. coliのラクトースパーミアーゼ(LacY)(J. Abramson et al., Science 301, 610 (Aug 1, 2003))およびE. coliのPi/グリセロール−3−ホスペート(phospate)(GlpT)(Y. Huang, M. J. Lemieux, J. Song, M. Auer, D. N. Wang, Science 301, 616 (Aug 1, 2003))の四次構造を共有していると考えられている。これらの例において、6つのN末端ヘリックスおよび6つのC末端へリックスは、へリックス6およびへリックス7の間にある細胞質内の長いループによって接続されている2つの異なるドメインを形成している。この対称性は、MFSに生じていると考えられる重複現象に対応している。基質は、N末端のドメインのへリックス1、2、4および5、ならびにC末端のドメインのへリックス7、8、10および11によって形成される親水性の空洞内に結合する。この空洞は、へリックス3、6、9および12によって安定化されている。
MFS(トランスポータ分類番号 2.A.1.1)の糖トランスポータファミリーは、膜貫通型のへリックス6および12(PESPR(配列番号9)/PETK(配列番号10))、ならびにループ2および8(GRR/GRK)に見られるモチーフによって特徴付けられている(M. C. Maiden, E. O. Davis, S. A. Baldwin, D. C. Moore, P. J. Henderson, Nature 325, 641 (Feb 12−18, 1987))。このファミリーとっての全体のHidden Markov Model(HMM)は、pfam.janelia.org/family/PF00083#tabview=tab3に見られ得る。PROSITE(N. Hulo et al., Nucleic Acids Res 34, D227 (Jan 1, 2006))は、このファミリーのメンバーを同定するために2つのモチーフを用いる。第1のモチーフは、[LIVMSTAG]−[LIVMFSAG]−“SH”−“RDE”−[LIVMSA]−[DE]−{TD}−[LIVMFYWA]−G−R−[RK]−x(4,6)−[GSTA](配列番号11)である。第2のモチーフは、[LIVMF]−x−G−[LIVMFA]−{V}−x−G−{KP}−x(7)−[LIFY]−x(2)−[EQ]−x(6)−[RK](配列番号12)である。PROSITEモチーフの読み方の例として、下記のモチーフ:[AC]−x−V−x(4)−{ED}は、[AlaまたはCys]−任意−Val−任意−任意−任意−任意−{GluまたはAspを除いて任意}(配列番号13)として解釈される。
本明細書に記載の通り、NCU00801、NCU00809、NCU08114、XP_001268541.1、およびLAC2は、セロデキストリンを輸送するポリペプチドをコードしていると見出された。さらに、アラニンスキャニング実験および配列解析を用いて、膜貫通型の12のα−へリックス、ならびに配列番号1、配列番号2、配列番号3、配列番号4、配列番号5、配列番号6、配列番号7、および配列番号8からなる群より選択される1つ以上の配列を含んでいる組換えポリペプチドが、セロデキストリンを輸送するポリペプチドをコードしていることを決定した。
したがって、一局面において、本発明は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでおり、膜貫通型のα−へリックス1は配列番号1を含んでいるポリぺプチドをコードしているポリヌクレオチドを提供する。他の局面において、本発明は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリぺプチドをコードしているポリヌクレオチドを提供する。ここで、膜貫通型のα−へリックス2は配列番号2を含んでいる。他の局面において、本発明は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリペプチドをコードしているポリヌクレオチドを提供する。ここで、膜貫通型のα−ヘリックス2および膜貫通型のα−ヘリックス3を接続しているループは配列番号3を含んでいる。他の局面において、本発明は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリぺプチドをコードしているポリヌクレオチドを提供する。ここで、膜貫通型のα−へリックス5は配列番号4を含んでいる。他の局面において、本発明は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリぺプチドをコードしているポリヌクレオチドを提供する。ここで、膜貫通型のα−へリックス6は配列番号5を含んでいる。他の局面において、本発明は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリぺプチドをコードしているポリヌクレオチドを提供する。ここで、膜貫通型のα−ヘリックス6および膜貫通型のα−へリックス7の間にある配列は配列番号6を含んでいる。他の局面において、本発明は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリぺプチドをコードしているポリヌクレオチドを提供する。ここで、膜貫通型のα−へリックス7は配列番号7を含んでいる。他の局面において、本発明は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリぺプチドをコードしているポリヌクレオチドを提供する。ここで、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11およびそれらの間の配列は配列番号8を含んでいる
上述の局面のそれぞれは、任意の数の組合せにおいて組み合わせられ得る。これらの局面のうちのいずれかに係るポリヌクレオチドは、配列番号1〜8のうちのいずれか1、2、3、4、5、6もしくは7つを含んでいるポリペプチド、または配列番号1〜8のすべてを含んでいるポリペプチドをコードし得る。例えば、ポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードし得、ここで、膜貫通型のα−ヘリックス1は配列番号1を含んでおり、膜貫通型のα−ヘリックス2および膜貫通型のα−ヘリックス3を接続しているループは配列番号3を含んでおり、膜貫通型のα−ヘリックス7は配列番号7を含んでいる。また、他の例において、ポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードし得、ここで、膜貫通型のα−ヘリックス2は配列番号2を含んでおり、膜貫通型のα−ヘリックス3は配列番号3を含んでおり、膜貫通型のα−ヘリックス6は配列番号5を含んでおり、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびこれらの間にある配列は配列番号8を含んでいる。
上述の局面のそれぞれは、任意の数の組合せにおいて組み合わせられ得る。これらの局面のうちのいずれかに係るポリヌクレオチドは、配列番号1〜8のうちのいずれか1、2、3、4、5、6もしくは7つを含んでいるポリペプチド、または配列番号1〜8のすべてを含んでいるポリペプチドをコードし得る。例えば、ポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードし得、ここで、膜貫通型のα−ヘリックス1は配列番号1を含んでおり、膜貫通型のα−ヘリックス2および膜貫通型のα−ヘリックス3を接続しているループは配列番号3を含んでおり、膜貫通型のα−ヘリックス7は配列番号7を含んでいる。また、他の例において、ポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードし得、ここで、膜貫通型のα−ヘリックス2は配列番号2を含んでおり、膜貫通型のα−ヘリックス3は配列番号3を含んでおり、膜貫通型のα−ヘリックス6は配列番号5を含んでおり、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびこれらの間にある配列は配列番号8を含んでいる。
上述の局面の一部の実施形態において、上記ポリペプチドは、少なくとも29%、少なくとも29%、少なくとも30%、少なくとも35%、少なくとも40%、少なくとも45%、少なくとも50%、少なくとも55%、少なくとも60%、少なくとも65%、少なくとも70%、少なくとも75%、少なくとも80%、少なくとも85%、少なくとも90%、少なくとも95%、少なくとも99%、または少なくとも100%の、NCU00801またはNCU08114に対するアミノ酸同一性をもつ。
本明細書にさらに記載されているように、NCU08221およびSTL12/XUT6は、キシロースを輸送するポリペプチドをコードしていることが見出された。XUT1は、アラビノースを輸送するポリペプチドをコードしていることが見出された。NCU06138は、アラビノースまたはグルコースを輸送するポリペプチドをコードしていることが見出された。SUT2、SUT3およびXUT3は、キシロースまたはグルコースを輸送するポリペプチドをコードしていることが見出された。NCU04963は、キシロース、アラビノース、またはグルコースを輸送するポリペプチドをコードしていることが見出された。好ましい実施形態において、本発明のポリヌクレオチドは、キシロースを輸送するNCU08221またはSTL12/XUT6であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる。他の好ましい実施形態において、本発明のポリヌクレオチドは、アラビノースを輸送するXUT1であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる。他の好ましい実施形態において、本発明のポリヌクレオチドは、グルコースを輸送するNCU06138であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる。他の好ましい実施形態において、本発明のポリヌクレオチドは、キシロースまたはグルコースを輸送するSUT2、SUT3、またはXUT3であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる。他の好ましい実施形態において、本発明のポリヌクレオチドは、キシロース、アラビノースまたはグルコースを輸送するNCU04963であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる。
NCU07705によってコードされているポリペプチドをコードしている本発明のポリヌクレオチドは、FunCat(Ruepp, 2004; webpage broad.mit.edu/annotation/genome/neurospora/Home.html)によって、未分類のタンパク質をコードしていると予想されている。しかし、NCU07705によってコードされている上記ポリペプチドのBLAST解析によって、当該ポリペプチドはC6ジンクフィンガードメインを含んでいる多くの転写因子と高い類似性を有していることが明らかになった。(図1を参照;例示的な相同物の一覧は、関連する米国特許出願第61/271,833号の図23に見られ得る)。本発明のポリヌクレオチドとしては、NCU0770によってコードされているポリペプチドのこれらの相同物、または当該分野に公知の任意の方法を用いて同定される任意の他の相同物をコードしているポリヌクレオチドが挙げられる。
本発明の他の局面において、本発明のポリペプチドは、NCU05137によってコードされているポリペプチドをコードしているポリヌクレオチドを含んでいる。NCU05137によってコードされているポリペプチドは、FunCatによって、未分類のタンパク質と分類されている。しかし、NCU05137は、多く糸状子嚢真菌のゲノムにおいて高度に保存されている(図2を参照)。本発明のポリヌクレオチドとしては、NCU05137によってコードされているポリペプチドの相同物または当該分野に公知の任意の方法を用いて同定される任意の他の相同物をコードしているポリヌクレオチドが挙げられる。
本発明の他の局面において、本発明のポリヌクレオチドは、NCU01517、NCU09133またはNCU10040によってコードされているポリペプチドをコードしているポリヌクレオチドを含んでいる。NCU01517によってコードされているポリペプチドは、FunCatによってグルコアミラーゼ前躯体と分類されている。NCU09133およびNCU10040によってコードされているポリペプチドは、FunCatによって未分類のタンパク質と分類されている。本発明のポリヌクレオチドとしては、NCU05137、NCU09133もしくはNCU10040によってコードされているポリペプチドの相同物、または当該分野に公知の任意の方法を用いて同定される任意の他の相同物をコードしているポリヌクレオチドが挙げられる。
これらのポリペプチドの推定される機能は、ポリヌクレオチドおよびそのコードされているタンパク質の機能的な分析を実施することによって確認され得る。これらの分析としては、例えば、上記ポリヌクレオチドの欠失を含んでいる株の表現型分析、遺伝学的な相補性実験、上記ポリヌクレオチドの野生型のコピーを過剰発現している株の表現型分析、上記ポリペプチドの組換え体の発現および精製、ならびにこれに続く組換えポリペプチドの生化学的な特性および活性の特徴付けが挙げられる。
本発明のポリヌクレオチドの配列は、当業者にとって公知の任意の好適な方法(例えば、直接の化学合成またはクローニングが挙げられる)によって調製される。直接の化学合成について、核酸の重合体の形成は、伸長するヌクレオチド鎖の末端5’水酸基に対する3’ブロックヌクレオチド単量体および5’ブロックヌクレオチド単量体の連続的な付加に典型的に関し、ここで、付加のそれぞれは、典型的に亜リン酸誘導体(例えば、リン酸トリエステルまたはホスホラミダイトなど)である付加された単量体の3’位に対する、伸長している鎖の末端5’水酸基の求核攻撃によってもたらされる。そのような方法論は、当業者にとって公知であり、関連する教科書および文献に記載されている(例えば、例えば、Matteucci et al., (1980) Tetrahedron Lett 21 : 719−722 ;米国特許第4,500,707号;米国特許第5,436,327号;および米国特許第5,700,637号)。さらに、所望される配列は、適切な制限酵素を用いてDNAを開裂させること、電気泳動を用いて断片を分離すること、その後に当業者にとって公知の方法(例えば、ポリメラーゼ連鎖反応(PCR;例えば、米国特許第4,683,195号))を介して所望の核酸配列をゲルから回収することによって、天然資源から単離され得る。
本発明のポリヌクレオチドは発現ベクターに組み込まれ得る。“発現ベクター”または“ベクター”は、宿主細胞に形質導入するか、宿主細胞を形質転換するか、または宿主細胞を感染させ、それによって細胞にとって本来のものではない核酸および/またはタンパクを発現させるか、または細胞にとって本来の様式ではない様式において核酸および/またはタンパクを発現させる化合物および/または組成物を指す。“発現ベクター”は、宿主細胞によって発現されるべき核酸の配列(通常はRNAまたはDNA)を含んでいる。また、発現ベクターは、宿主細胞への核酸の侵入を補助する物質(例えば、ウイルス、リポソーム、またはタンパク質外被など)を、必要に応じて含んでいる。本発明における用途について考慮される発現ベクターとしては、任意の好ましい作動性エレメントまたは任意の必要な作動性エレメントとともに、核酸が挿入され得るベクターが挙げられる。さらに、発現ベクターは宿主細胞に移送され得、宿主細胞において複製され得るベクターである必要がある。好ましい発現ベクターは、プラスミド、特に十分に実証されており、核酸配列の転写にとって好ましいか、または必要な作動性エレメントを含んでいる制限酵素認識部位を有しているプラスミドである。そのようなプラスミドおよび他の発現ベクターは、当業者にとって周知である。
個別のポリヌクレオチドの組込みは、公知の方法(発現ベクター(例えばプラスミド)の特定の部位を切断するための制限酵素(例えば、BamHI、EcoRI、HhaI、XhoIおよびXmaIなど)の使用が挙げられる)によって達成され得る。制限酵素は、切断された発現ベクターの末端に対して相補的な配列を有しているポリヌクレオチドとアニールされ得るか、当該配列を有するために合成され得る一本鎖の末端をもたらす。アニーリングは適切な酵素(例えばDNAリガーゼ)を用いて行われる。当業者によって適切に理解される通り、発現ベクターおよび所望のポリヌクレオチドの両方は、同じ制限酵素を用いてしばしば切断され、これによって当該発現ベクターの末端および当該ポリヌクレオチドの末端が互いに相補的であることを保証する。さらに、DNAリンカーは、発現ベクターへの核酸配列の連結を容易にするために使用され得る。
また、一組の個別のポリヌクレオチドは、当業者にとって公知の方法を利用することによって組み合せられ得る(例えば、米国特許第4,683,195号)。
例えば、所望のポリヌクレオチドのそれぞれは、別々のPCRにおいてまず生成され得る。その後、特異的なプライマーは、PCR産物の末端が相補的な配列を含んでいるように設計される。上記PCR産物が混合され、変性され、ふたたびアニールされるとき、それらの3’末端において一致配列を有している鎖は、重なり合い、互いにプライマーとして機能し得る。DNAポリメラーゼによるこの重複の伸長は、元の配列がともに“スプライス”されている分子を生成する。この方法において、一組の個別のポリヌクレオチドは、ともに“スプライス”されており、続いて同時に宿主細胞に形質導入される。したがって、複数のポリヌクレオチドのそれぞれの発現は、関連付けられている。
それから、個別のポリヌクレオチドまたは“スプライス”されたポリヌクレオチドは、発現ベクターに組み込まれる。本発明は、ポリヌクレオチドが発現ベクターに組み込まれる過程について限定されない。当業者は、発現ベクターにポリヌクレオチドを組み込むために必須の工程に精通している。典型的な発現ベクターは、リボソーム結合部位とともに1つ以上の制御領域(例えば、E. coliにおいて開始コドンの3〜11ヌクレオチド上流が配置されている3〜9ヌクレオチドの長さのヌクレオチド配列)が上流に存在している所望のポリヌクレオチドの配列を含んでいる。Shine and Dalgarno (1975) Nature 254(5495):34-38およびSteitz (1979) Biological Regulation and Development (ed. Goldberger, R. F.), 1:349 - 399 (Plenum, New York)を参照すればよい。
本明細書に使用されるとき、“作動可能に連結されている”という用語は、制御配列が、DNA配列のコーディング配列、または当該制御配列がポリペプチドの発現を指揮するようなポリヌクレオチドに対する適切な位置に配置されている構成を指す。
制御領域としては、例えばプロモータおよびオペレータを含んでいる領域が挙げられる。プロモータは、所望のポリヌクレオチドに対して作動可能に連結されており、これによって、RNAポリメラーゼ酵素を介してポリヌクレオチドの転写を開始させる。オペレータは、抑制因子タンパク質が結合し得るタンパク質結合ドメインを含んでいる、プロモータに隣接している核酸の配列である。抑制因子タンパク質の非存在下において、転写はプロモータを介して開始する。抑制因子タンパク質が存在しているとき、上記オペレータのタンパク質結合ドメインに特異的な抑制因子タンパク質が当該オペレータに結合し、これによって転写を阻害する。この方法によって、使用される特定の制御領域および対応する抑制因子タンパク質の存在または非存在に基づいて、転写の制御が達成される。例としては、ラクトースプロモータ(ラクトースと接触しているときにLad抑制因子タンパク質は構造を変化させ、これによって、Lad抑制因子タンパク質がオペレータに結合することを防止する)、およびトリプトファンプロモータ(トリプトファンと複合化されているときに、TrpR抑制因子タンパク質はオペレータと結合しない立体配座を有している)が挙げられる。他の例は、tacプロモータである(de Boer et al., (1983) Proc Natl Acad Sci USA 80 (1) : 21-25を参照)。当業者によって適切に理解される通り、これらの発現ベクターおよび他の発現ベクターは、本発明において使用され得、本発明はこの点について限定されない。
任意の好適な発現ベクターは、所望の配列を組み込むために使用され得るが、容易に入手可能な発現ベクターとしては、限定されることなく、プラスミド(例えば、pSClOl、pBR322、pBBRlMCS−3、pUR、pEX、pMRlOO、pCR4、pBAD24、pUC19);ファージ(例えば、Ml 3ファージおよびλファージ)が挙げられる。もちろん、そのような発現ベクターは、特定の宿主細胞にとってのみ好適であり得る。しかし、当業者は、任意の特定の発現ベクターが所定の任意の宿主細胞に適しているか否かをどうか、通常の実験によって容易に決定し得る。例えば、発現ベクターは宿主細胞に導入され、それから当該宿主細胞は、生存能力およびベクターに含まれている配列の発現について観察される。さらに、発現ベクターおよび任意の特定の宿主細胞に対するそれらの適合性について説明している関連する教科書および文献が参照され得る。
〔本発明の宿主細胞〕
本明細書における発明は、セロデキストリンまたはペントースを細胞に輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞に関する。本明細書にさらに記載されているのは、宿主細胞へのセロデキストリンの輸送を促進させる方法、セロデキストリンを含んでいる培地における宿主細胞の成長を促進させる方法、セルロース由来およびヘミセルロース由来の糖を共発酵する方法、ならびにセロデキストリンを細胞に輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備することによって、炭化水素または炭化水素の誘導体を製造する方法である。本明細書にさらに記載されているのは、宿主細胞へのペントースの輸送を促進させる方法、ペントースを含んでいる培地における宿主細胞の成長を促進させる方法、およびペントースを細胞に輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備することによって、炭化水素または炭化水素の誘導体を製造する方法である。
本明細書における発明は、セロデキストリンまたはペントースを細胞に輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞に関する。本明細書にさらに記載されているのは、宿主細胞へのセロデキストリンの輸送を促進させる方法、セロデキストリンを含んでいる培地における宿主細胞の成長を促進させる方法、セルロース由来およびヘミセルロース由来の糖を共発酵する方法、ならびにセロデキストリンを細胞に輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備することによって、炭化水素または炭化水素の誘導体を製造する方法である。本明細書にさらに記載されているのは、宿主細胞へのペントースの輸送を促進させる方法、ペントースを含んでいる培地における宿主細胞の成長を促進させる方法、およびペントースを細胞に輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備することによって、炭化水素または炭化水素の誘導体を製造する方法である。
“宿主細胞”および“宿主微生物”は、組換えのDNAまたはRNAの挿入を介して形質転換され得る生体の生物学的な細胞を指して、本明細書において交換可能に使用される。そのようなDNAまたはRNAは発現ベクターに存在し得る。したがって、本明細書における宿主生物または細胞は、原核生物(例えば、Eubacteria界)または真核細胞であり得る。当業者によって適切に理解される通り、真核細胞は膜に区切られている核を有しているが、原核細胞は膜に区切られている核を欠いている。
任意の原核宿主細胞または真核宿主細胞は、核酸の配列を用いて形質転換された後に生存性を維持する限り、本発明に使用され得る。宿主細胞は、必要な核酸配列の形質導入、タンパク質(例えばトランスポータ)または中間生成物のそれに続く発現に悪影響を及ぼさないことが好ましい。好適な真核細胞としては、真菌、植物、昆虫、または動物細胞が挙げられるが、これらに限定されない。
好ましい実施形態において宿主細胞は真菌株である。本明細書に使用されるとき、“真菌”は、phyla Ascomycota、Basidiomycota、ChytridiomycotaおよびZygomycota(Hawksworth et al., Ainsworth and Bisby's Dictionary of The Fungi, 8th edition, 1995, CAB International, University Press, Cambridge, UKに定義付けられているような)、ならびにOomycota(Hawksworth et al., 1995, supra, page 171に挙げられているような)およびすべての栄養胞子形成菌(Hawksworth et al., 1995, supra)を包含している。
一部の実施形態において、真菌の宿主は酵母株である。本明細書に使用されるとき、“酵母”は、有子嚢胞子酵母(エンドマイセス属)、担子胞子酵母、および不完全真菌(分芽菌属)に属する酵母を包含している。酵母の分類は将来的に変化し得るので、本発明の目的のために、酵母はBiology and Activities of Yeast(Skinner, F. A., Passmore, S. M., and Davenport, R. R., eds, Soc. App. Bacteriol. Symposium Series No. 9, 1980)に記載の通りに定義付けられる。
より好ましい実施形態において、酵母宿主は、Candida、Hansenula、Kluyveromyces、Pichia、Saccharomyces、Schizosaccharomyces、またはYarrowia株である。
一部の実施形態において、酵母の宿主は、Saccharomyces carlsbergensis株(Todkar, 2010)、Saccharomyces cerevisiae株(Duarte et al., 2009)、Saccharomyces diastaticus株、Saccharomyces douglasii株、Saccharomyces kluyveri株、Saccharomyces norbensis株、Saccharomyces monacensis株(GB−Analysts Reports, 2008)株、Saccharomyces bayanus株(Kristen Publicover, 2010)、Saccharomyces pastorianus株(Nakao et al., 2007)、Saccharomyces pombe株(Mousdale, 2008)、またはSaccharomyces oviformis株である。他の好ましい実施形態において、酵母の宿主は、Kluyveromyces lactis(O.W. Merten, 2001)、Kluyveromyces fragilis(Pestal et al., 2006; Siso, 1996)、Kluyveromyces marxiamus(K. Kourkoutas et al., 2008)、Pichia stipitis(Almeida et al., 2008)、Candida shehatae(Ayhan Demirbas, 2003)、または Candida tropicalis(Jamai et al., 2006)である。他の実施形態において、酵母の宿主は、Yarrowia lipolytica(Biryukova E.N., 2009)、Brettanomyces custersii(Spindler D.D. et al., 1992)、またはZygosaccharomyces roux(Chaabane et al., 2006)であり得る。
他の一部の実施形態において、真菌の宿主は糸状菌株である。“糸状菌”は、亜門であるEumycotaおよびOomycotaのすべての糸状の形態を包含している(上述のHawksworth et al., 1995によって定義付けられているように)。糸状菌は、一般的に、キチン、セルロース、グルカン、キトサン、マンナンおよび複合的な他の多糖によって構成されている菌糸体の壁によって一般的に特徴付けられている。栄養生長は菌糸の伸長によるものであり、炭素異化は必須に好気性である。対照的に、酵母(例えばSaccharomyces cerevisiae)による栄養生長は、単細胞の葉状体の出芽によるものであり、炭素異化は発酵性であり得る。
好ましい実施形態において、糸状菌宿主は、Acremonium株、Aspergillus株、Fusarium株、Humicola株、Mucor株、Myceliophthora株、Neurospora株、Penicillium株、Scytalidium株、Thielavia株、Tolypocladium株、またはTrichoderma株であるが、これらに限定されない。
一部の実施形態において、糸状菌宿主は、Aspergillus awamori株、Aspergillus foetidus株、Aspergillus japonicus株、Aspergillus nidulans株、Aspergillus niger株または Aspergillus oryzae株である。他の実施形態において、糸状菌宿主は、Fusarium bactridioides株、Fusarium cerealis株、Fusarium crookwellense株、Fusarium culmorum株、Fusarium graminearum株、Fusarium graminum株、Fusarium heterosporum株、Fusarium negundi株、Fusarium oxysporum株、Fusarium reticulatum株、Fusarium roseum株、Fusarium sambucinum株、Fusarium sarcochroum株、Fusarium sporotrichioides株、Fusarium sulphureum株、Fusarium torulosum株、Fusarium trichothecioides株、またはFusarium venenatum株である。さらに好ましい他の実施形態において、糸状菌宿主は、Humicola insolens株、Humicola lanuginosa株、Mucor miehei株、Myceliophthora thermophila株、Neurospora crassa株、Penicillium purpurogenum株、Scytalidium thermophilum株、Sporotrichum thermophile株(Topakas et al., 2003)、またはThielavia terrestris株である。さらなる実施形態において、糸状菌宿主は、Trichoderma harzianum株、Trichoderma koningii株、Trichoderma longibrachiatum株、Trichoderma reesei株、またはTrichoderma viride株である。
他の好ましい実施形態において、宿主細胞は原核生物であり、一部の実施形態において、原核生物はE. coli(Dien, B.S. et al., 2003; Yomano, L. P. et al., 1998; Moniruzzaman et al., 1996)、Bacillus subtilis(Susana Romero et al., 2007)、Zymomonas mobilis(B. S. Dien et al, 2003; Weuster Botz, 1993; Alterthum and Ingram, 1989)、Clostridium sp.(Zeikus, 1980; Lynd et al., 2002; Demain et al., 2005)、Clostridium phytofermentans(Leschine S., 2010)、Clostridium thermocellum (Lynd et al., 2002)、Clostridium beijerinckii(Giles Clark, 2008)、Clostridium acetobutylicum(Moorella thermoacetica)(Huang W.C. et al., 2004; Dominik et al., 2007)、Thermoanaerobacterium saccharolyticum(Marietta Smith, 2009)、またはKlebsiella oxytoca(Dien, B.S. et al., 2003; Zhou et al., 2001; Brooks およびIngram, 1995)である。他の実施形態において、原核宿主細胞は、Carboxydocella sp.(Dominik et al., 2007)、またはCorynebacterium glutamicum (Masayuki Inui, et al., 2004)、Enterobacteriaceae(Ingram et al., 1995)、Erwinia chrysanthemi (Zhou および Ingram, 2000; Zhou et al., 2001)、Lactobacillus sp. (McCaskey, T.A., et al., 1994)、Pediococcus acidilactici(Zhou, S. et al., 2003)、Rhodopseudomonas capsulata(X.Y. Shi et al., 2004)、Streptococcus lactis(J.C. Tang et al., 1988)、Vibrio furnissii(L.P. Wackett, 2010)、Vibrio furnissii M1 (Park et al, 2001)、Caldicellulosiruptまたは saccharolyticus (Z. Kadar et al., 2004)、または Xanthomonas campestris(S.T. Yang et al., 1987)である。他の実施形態において、宿主細胞はシアノバクテリアである。細菌性の宿主細胞のさらなる例としては、限定されることなく。Escherichia、Enterobacter、Azotobacter、Erwinia、Bacillus、Pseudomonas、Klebsiella、Proteus、Salmonella、Serratia、Shigella、Rhizobia、Vitreoscilla、Synechococcus、Synechocystis、およびParacoccusの分類学的な分類に割り当てられている種が挙げられる。
本発明の特に好ましい実施形態において、宿主細胞は、Saccharomyces sp.、Saccharomyces cerevisiae、Saccharomyces monacensis、Saccharomyces bayanus、Saccharomyces pastorianus、Saccharomyces carlsbergensis、Saccharomyces pombe、Kluyveromyces sp.、Kluyveromyces marxiamus、Kluyveromyces lactis、Kluyveromyces fragilis、Pichia stipitis、Sporotrichum thermophile、Candida shehatae、Candida tropicalis、Neurospora crassa、Zymomonas mobilis、Clostridium sp.、Clostridium phytofermentans、Clostridium thermocellum、Clostridium beijerinckii、Clostridium acetobutylicum、Moorella thermoacetica、Escherichia coli、Klebsiella oxytoca、Thermoanaerobacterium saccharolyticum、または Bacillus subtilisである。Saccharomyces sp.としては工業的なSaccharomyces株が挙げられる。Argueso et al.は、バイオエタノール産生に一般的に使用される工業的なSaccharomyces株のゲノム構造、およびバイオエタノール産生に重要な特定の遺伝子多型について議論している(Genome Research, 19: 2258−2270, 2009)。
本発明の宿主細胞は、組換え核酸が宿主細胞に導入されて、遺伝学的に変更され得る。したがって、そのような遺伝的に変更された宿主細胞は天然に存在しない。好適な宿主細胞は、機能について異なっている1つ以上のタンパク質をコードしている1つ以上の核酸コンストラクトを発現可能な細胞である。
本明細書に使用されるとき、“組換え核酸”または“異種核酸”または“組換えポリヌクレオチド”は、以下の少なくとも1つ:(a)所定の宿主細胞にとって核酸の配列が外来性である(すなわち、天然には細胞に見られない);(b)配列は所定の宿主細胞において天然に見られ得るが、異常な(例えば予想を超えた)量において認められ得る;または(c)核酸の配列は互いに同じ関係において天然に見られない2つ以上の部分配列を含んでいる、を満たしている核酸の重合体を指す。例えば、例(c)に関して、組換え核酸配列は、新たな機能的な核酸を作製ために配置されている無関係な遺伝子に由来する2つ以上の配列を有している。特に、本発明は、宿主細胞への発現ベクターの導入について説明しており、当該発現ベクターは、宿主細胞において通常には見られないタンパク質をコードしている核酸配列を含んでいるか、または細胞において通常に見られるが、異なる制御配列の制御下にあるタンパク質をコードしている核酸配列を含んでいる。その結果として、宿主細胞のゲノムに関して、タンパク質をコードしている核酸配列は組換え体である。
いくつかの実施形態において、宿主細胞は、本発明のポリヌクレオチドによってコードされている任意のタンパク質を必然的に生成する。所望のタンパク質をコードしている遺伝子は宿主細胞にとって異種であり得るか、またはこれらの遺伝子は宿主細胞にとって内因性であり得るが、宿主細胞の遺伝子のさらなる高発現をもたらす異種のプロモータおよび/または制御領域と作動可能に連結されている。他の実施形態において、宿主細胞は、所望のタンパク質を自然には生成せず、それらの分子の生成に必要な1つ以上の遺伝子の発現が可能な異種の核酸コンストラクトを含んでいる。
核酸分子もしくはポリペプチドおよび特定の細胞もしくは微生物に言及して本明細書に使用されるとき、“内因性”は、組換え改変技術を用いて細胞内に導入されなかった、細胞に存在している核酸配列またはペプチド(例えば、細胞が天然から最初に単離されたときに、細胞に存在した遺伝子)を指す。
“遺伝学的に改変された”または“遺伝学的に変更された”は、上昇したレベル、低下したレベル、または変異された形態においてタンパク質を発現する原核宿主細胞または真核宿主細胞を作製するために用いられる任意の組換えDNA法もしくは組換えRNA法を指す。言い換えると、宿主細胞は、組換えポリヌクレオチド分子を用いて形質移入されるか、形質転換されるか、または形質導入され、それによって細胞が所望のタンパク質の発現を変更するように変えられている。宿主細胞を遺伝学的に改変させる方法およびベクターは、当業者によく知られている;例えば種々の技術が、Current Protocols in Molecular Biology, Ausubel et al., eds.(Wiley & Sons, New York, 1988, and quarterly updates)に示されている。遺伝学的改変技術としては、発現ベクター、標的化された相同性組換えおよび遺伝子活性化(例えば、Chappelの米国特許第5,272,071号を参照)、ならびに改変された転写因子によるトランス活性化(例えば、, Segal et al., (1999) Proc Natl Acad Sci USA 96(6):2758−2763参照)が挙げられるが、これらに限定されない
遺伝子の発現または機能の促進をもたらす遺伝子の修飾は、遺伝子の増幅、過剰生産、過剰発現、活性化、促進、増強、付加または上方制御と呼ばれ得る。より詳細には、本明細書において述べられている酵素または他のタンパク質の作用(または活性)を促進することに対する言及は、酵素またはタンパク質の促進された発現および/または機能性(生物学的な活性)をもたらす、該当する宿主細胞の遺伝学的な任意の修飾を指し、タンパク質のより高い活性もしくは作用(例えば、特異的な活性または生体内の酵素活性)、タンパク質の低下した阻害もしくは分解、およびタンパク質の過剰発現を包含している。例えば、遺伝子のコピー数が増加され得るか、天然のプロモータと比べて高い発現レベルをもたらすプロモータの使用によって発現レベルは増加され得るか、または遺伝子は、遺伝的な改変もしくは古典的な変異生成によって変更されて、酵素の生物学的な活性もしくはタンパク質の作用を増強させ得る。また、これらの修飾のうちいくつかの組合せが可能である。
遺伝子の発現または機能の促進をもたらす遺伝子の修飾は、遺伝子の増幅、過剰生産、過剰発現、活性化、促進、増強、付加または上方制御と呼ばれ得る。より詳細には、本明細書において述べられている酵素または他のタンパク質の作用(または活性)を促進することに対する言及は、酵素またはタンパク質の促進された発現および/または機能性(生物学的な活性)をもたらす、該当する宿主細胞の遺伝学的な任意の修飾を指し、タンパク質のより高い活性もしくは作用(例えば、特異的な活性または生体内の酵素活性)、タンパク質の低下した阻害もしくは分解、およびタンパク質の過剰発現を包含している。例えば、遺伝子のコピー数が増加され得るか、天然のプロモータと比べて高い発現レベルをもたらすプロモータの使用によって発現レベルは増加され得るか、または遺伝子は、遺伝的な改変もしくは古典的な変異生成によって変更されて、酵素の生物学的な活性もしくはタンパク質の作用を増強させ得る。また、これらの修飾のうちいくつかの組合せが可能である。
遺伝子の発現、遺伝子の機能、または遺伝子産物(すなわち、遺伝子によってコードされているタンパク質)の機能の抑制をもたらす遺伝子の修飾は、不活性化(完全または部分的な)、欠失、中断、妨害、サイレンシング、または下方制御(すなわち遺伝子の発現の減弱)を指し得る。例えば、遺伝子によってコードされているタンパク質の機能の低下をもたらす遺伝子の遺伝学的な修飾は、遺伝子の完全な欠失(すなわち、遺伝子が存在せず、したがってタンパク質が存在しない)、タンパク質の不完全な翻訳もしくは翻訳の消失(例えばタンパク質は発現されない)をもたらす遺伝子の変異、またはタンパク質の天然の機能を低下させるか、もしくは消失させる(例えば、低下しているかもしくは消失している酵素活性もしくは作用を有しているタンパク質が発現される)遺伝子の変異の結果であり得る。より詳細には、本明細書に述べられているタンパク質の活性の低下に対する言及は、タンパク質の低下した発現および/または機能性(生物学な活性)をもたらす、該当の宿主細胞の任意の遺伝学的な修飾を一般的に指しており、タンパク質の発現の低下または消失と同時に、タンパク質の低下した活性(例えば低下した輸送)、タンパク質の増強された阻害または分解を包含している。例えば、本発明のタンパク質の作用または活性は、タンパク質の産生を阻害こともしくは低下させること、タンパク質の作用を低下させること、またはタンパク質の作用を抑制することによって低下させられ得る。また、これらの修飾のうちいくつかの組合せが可能である。タンパク質の産生を阻害することまたは低減させることは、成長培地における誘導性化合物の存在を要求するプロモータの調節下に、タンパク質をコードしている遺伝子を配置することを包含し得る。誘導物質が培地から枯渇するような条件を確立することによって、タンパク質をコードしている遺伝子の発現(したがってタンパク質合成)は停止され得る。また、タンパク質の作用を阻害することまたは低下させることは、参照によって本明細書に援用される米国特許第4,743,546号に記載のような切除技術手法を用いることを包含し得る。この手法を用いるために、対象のタンパク質をコードしている遺伝子は、ゲノムからの上記遺伝子の特異的な調節された切除を可能にする、特異的な遺伝学的な配列の間にクローン化される。切除は、例えば、米国特許第4,743,546号にあるような培養の培養温度の変更、またはいくつかの他の物理的シグナルもしくは栄養性のシグナルによって促進され得る。
一般的に、本発明によれば、変異タンパク質または修飾タンパク質の所定の特性(例えば酵素活性、化合物の輸送能)の向上または低下は、同一または同等の条件において測定されるか、または明らかにされる、同じ生物(同一の起源または親配列)に由来する野生型の(すなわち改変されていない通常の)タンパク質の同じ特性を基準にしてなされている。同様に、遺伝学的に修飾された宿主細胞の性質(例えばタンパク質の発現および/または生物学的な活性、または生成物の生成)の向上または低下は、同一または同等の条件におけるの同じ種、好ましくは同じ株の野生型の宿主細胞の同じ性質を基準にしてなされている。そのような条件としては、タンパク質の活性(例えば発現または生物学的な活性)または宿主細胞の他の性質が測定されるアッセイまたは培養の条件(例えば培地の成分、温度、pHなど)、ならびに用いられるアッセイの種類、評価される宿主細胞などが挙げられる。上述のように、同等の条件は、同じ条件においてなされる比較に照らして、細胞増殖、酵素の発現または生物学的な活性に対する影響を実質的に変更させない、必ずしも同一ではないが、類似する(例えばいくつかの保存的な条件の変化が許容され得る)条件(例えば培養条件)である。
所定のタンパク質(例えばトランスポータ、酵素)の活性を向上させるか、または低下させる遺伝学的な修飾を有している遺伝学的に変更された宿主細胞は、野生型の宿主細胞における野生型のタンパク質の活性と比較して、少なくとも5%、およびより好ましくは少なくとも約10%、より好ましくは少なくとも約15%、より好ましくは少なくとも約20%、より好ましくは少なくとも約25%、より好ましくは少なくとも約30%、より好ましくは少なくとも約35%、より好ましくは少なくとも約40%、より好ましくは少なくとも約45%、より好ましくは少なくとも約50%、より好ましくは少なくとも約55%、より好ましくは少なくとも約60%、より好ましくは少なくとも約65%、より好ましくは少なくとも約70%、より好ましくは少なくとも約75%、より好ましくは少なくとも約80%、より好ましくは少なくとも約85%、より好ましくは少なくとも約90%、より好ましくは少なくとも約95%、または5%〜100%の間にある全整数における任意のパーセンテージ(例えば6%、7%、8%など)の、タンパク質の活性または作用(例えば発現、産生および/または生物学的な活性)の向上または低下をそれぞれ示す。単離された修飾核酸分子または修飾タンパク質を、単離された野生型の核酸またはタンパク質と直接的に比較する場合(例えば比較がインビボと比べてインビボに対してなされる場合)、同じ差異が好ましい。
本発明の他の局面において、所定のタンパク質(例えばトランスポータ、酵素)の活性を向上させるか、または低下させる遺伝学的な修飾を有している遺伝学的に変更された宿主細胞は、野生型の宿主細胞における野生型タンパク質の活性と比べて、少なくとも約2倍、より好ましくは少なくとも約5倍、より好ましくは少なくとも約10倍、より好ましくは少なくとも約20倍、より好ましくは少なくとも約30倍、より好ましくは少なくとも約40倍、より好ましくは少なくとも約50倍、より好ましくは少なくとも約75倍、より好ましくは少なくとも約100倍、より好ましくは少なくとも約125倍、より好ましくは少なくとも約150倍、または少なくとも約2倍から始まる任意の全整数の増加(例えば3倍、4倍、5倍、6倍など)の、タンパク質の活性または作用(例えば発現、生産および/または生物学的な活性)の向上または低下をそれぞれ示す。
(宿主細胞の構成要素)
一局面において、本発明の宿主細胞は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリペプチドをコードしているポリヌクレオチドを含んでいる。ここで、膜貫通型のα−へリックス1は配列番号1を含んでいる。他の局面において、本発明の宿主細胞は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリペプチドをコードしているポリヌクレオチドを含んでいる。ここで、膜貫通型のα−へリックス2は配列番号2を含んでいる。他の局面において、本発明の宿主細胞は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリペプチドをコードしているポリヌクレオチドを含んでいる。膜貫通型のα−ヘリックス2および膜貫通型のα−ヘリックス3を接続しているループは配列番号3を含んでいる。他の局面において、本発明の宿主細胞は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリペプチドをコードしているポリヌクレオチドを含んでいる。ここで、膜貫通型のα−ヘリックス5は配列番号4を含んでいる。他の局面において、本発明の宿主細胞は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでおり、膜貫通型のα−ヘリックス6は配列番号5を含んでいるポリペプチドをコードしているポリヌクレオチドを含んでいる。他の局面において、本発明の宿主細胞は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリペプチドをコードしているポリヌクレオチドを含んでいる。ここで、膜貫通型のα−ヘリックス6および膜貫通型のα−ヘリックス7の間にある配列は配列番号6を含んでいる。他の局面において、本発明の宿主細胞は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリペプチドをコードしているポリヌクレオチドを含んでいる。ここで、膜貫通型のα−ヘリックス7は配列番号7を含んでいる。他の局面において、本発明の宿主細胞は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリペプチドをコードしているポリヌクレオチドを含んでいる。ここで、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびそれらの間にある配列は配列番号8を含んでいる。
一局面において、本発明の宿主細胞は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリペプチドをコードしているポリヌクレオチドを含んでいる。ここで、膜貫通型のα−へリックス1は配列番号1を含んでいる。他の局面において、本発明の宿主細胞は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリペプチドをコードしているポリヌクレオチドを含んでいる。ここで、膜貫通型のα−へリックス2は配列番号2を含んでいる。他の局面において、本発明の宿主細胞は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリペプチドをコードしているポリヌクレオチドを含んでいる。膜貫通型のα−ヘリックス2および膜貫通型のα−ヘリックス3を接続しているループは配列番号3を含んでいる。他の局面において、本発明の宿主細胞は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリペプチドをコードしているポリヌクレオチドを含んでいる。ここで、膜貫通型のα−ヘリックス5は配列番号4を含んでいる。他の局面において、本発明の宿主細胞は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでおり、膜貫通型のα−ヘリックス6は配列番号5を含んでいるポリペプチドをコードしているポリヌクレオチドを含んでいる。他の局面において、本発明の宿主細胞は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリペプチドをコードしているポリヌクレオチドを含んでいる。ここで、膜貫通型のα−ヘリックス6および膜貫通型のα−ヘリックス7の間にある配列は配列番号6を含んでいる。他の局面において、本発明の宿主細胞は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリペプチドをコードしているポリヌクレオチドを含んでいる。ここで、膜貫通型のα−ヘリックス7は配列番号7を含んでいる。他の局面において、本発明の宿主細胞は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリペプチドをコードしているポリヌクレオチドを含んでいる。ここで、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびそれらの間にある配列は配列番号8を含んでいる。
上述の局面のそれぞれは、任意の数の組合せにおいて組み合わせられ得る。宿主細胞は、配列番号1〜8のうちのいずれか1、2、3、4、5、6または7つの配列を含んでいるポリペプチド、または配列番号1〜8のすべての配列を含み得るポリペプチドをコードしているポリヌクレオチドを含み得る。例えば、宿主細胞は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリペプチドをコードしているポリヌクレオチドを含み得る。ここで、膜貫通型のα−ヘリックス1は配列番号1を含んでおり、膜貫通型のα−ヘリックス2および膜貫通型のα−ヘリックス3を接続しているループは配列番号3を含んでおり、膜貫通型のα−ヘリックス7は配列番号7を含んでいる。他の例において、宿主細胞は、膜貫通型のα−へリックス1、膜貫通型のα−へリックス2、膜貫通型のα−へリックス3、膜貫通型のα−へリックス4、膜貫通型のα−へリックス5、膜貫通型のα−へリックス6、膜貫通型のα−へリックス7、膜貫通型のα−へリックス8、膜貫通型のα−へリックス9、膜貫通型のα−へリックス10、膜貫通型のα−へリックス11、膜貫通型のα−へリックス12を含んでいるポリペプチドをコードしているポリヌクレオチドを含み得る。ここで、膜貫通型のα−へリックス2は配列番号2を含んでおり、膜貫通型のα−ヘリックス3は配列番号3を含んでおり、α−ヘリックス6は配列番号5を含んでおり、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびこれらの間にある配列は配列番号8を含んでいる。
上述の局面の一部の実施形態において、上記ポリペプチドは、少なくとも29%、少なくとも30%、少なくとも35%、少なくとも40%、少なくとも45%、少なくとも50%、少なくとも55%、少なくとも60%、少なくとも65%、少なくとも70%、少なくとも75%、少なくとも80%、少なくとも85%、少なくとも90%、少なくとも95%、少なくとも99%、または少なくとも100%の、NCU00801またはNCU08114に対するアミノ酸同一性を有している。
好ましい実施形態において、上記宿主細胞は、β−グルコシダーゼの触媒作用ドメインをコードしているポリヌクレオチドをさらに含んでいる。本明細書に使用されるとき、β−グルコシダーゼは、β−D−グルコースの放出にともなう、末端の非還元性のβ−D−グルコース残基の加水分解を触媒するβ−D−グルコシドグルコヒドロラーゼ(E.C.3.2.1.21)を指す。β−グルコシダーゼの触媒作用ドメインは、例えばVenturi et al., 2002.によって説明されている基礎的な手法にしたがって、決定されるようなβ−グルコシダーゼ活性を有している。β−グルコシダーゼの触媒作用ドメインは、β−D−グルコースの遊離をともなう、末端の非還元性のβ−D−グルコース残基の加水分解を触媒する任意の領域である。好ましい実施形態において、β−グルコシダーゼはグリコシルヒドロラーゼファミリー1のメンバーである。このグループのメンバーは、モチーフ[LIVMFSTC]−[LIVFYS]−[LIV]−[LIVMST]−E−N−G−[LIVMFAR]−[CSAGN](配列番号14)によって同定され得る。ここで、Eは触媒作用性のあるグルタミン酸である (ウェブページexpasy.org/cgi−bin/prosite−search−ac?PDOC00495)。一部の実施形態において、β−グルコシダーゼの触媒作用ドメインをコードしているポリヌクレオチドは宿主細胞にとって異種である。好ましい実施形態において、β−グルコシダーゼの触媒作用ドメインは宿主細胞において細胞内に局在している。好ましい実施形態において、β−グルコシダーゼはN. crassaに由来し、特に好ましい実施形態において、β−グルコシダーゼはNCU00130である。一部の実施形態において、β−グルコシダーゼはNCU00130の相同分子種であり得る。NCU00130の相同分子種の例としては、限定されることなく、T. melanosporum、CAZ82985.1;A. oryzae、BAE57671.1;P. placenta、EED81359.1;P. chrysosporium、BAE87009.1;Kluyveromyces lactis、CAG99696.1;Laccaria bicolor、EDR09330;Clavispora lusitaniae、EEQ37997.1;およびPichia stipitis、ABN67130.1が挙げられる。使用され得る他のβ−グルコシダーゼとしては、グリコシルヒドロラーゼファミリー3に由来するβ−グルコシダーゼが挙げられる。これらのβ−グルコシダーゼは、PROSITEにしたがってモチーフ:[LIVM](2)−[KR]−x−[EQKRD]−x(4)−G−[LIVMFTC]−[LIVT]−[LIVMF]−[ST]−D−x(2)−[SGADNIT](配列番号15)によって同定され得る。したがって、Dは触媒作用性のアスパラギン酸である。NCBI配列COG2723に見られるβ−グルコシダーゼ/6−ホスホ−β−グルコシダーゼ/β−グルコシダーゼの保存された領域を含んでいる任意のβ−グルコシダーゼが、使用され得る。特定のβ−グルコシダーゼから得られた触媒作用領域は、宿主細胞に含まれているセロデキストリントランスポータに依存して選択され得る。
一部の実施形態において、宿主細胞は、ペントース利用に関与する1つ以上の酵素をコードしている1つ以上のポリヌクレオチドを含んでいる。1つ以上のポリヌクレオチドは内因性または宿主細胞にとって異種であり得る。本明細書に使用されるとき、ペントースは5つの炭素源子を有している単糖を指す。ペントースの例としては、限定されることなく、キシロース、アラビノース、マンノース、ガラクトース、およびラムノースが挙げられる。ペントース利用に関与する1つ以上の酵素は、限定されることなく、例えば、L−アラビノースイソメラーゼ、L−リブロキナーゼ、L−リブロース−5−P 4 エピメラーゼ、キシロースイソメラーゼ、キシルロキナーゼ、アルドースリダクターゼ、L−アラビニトール 4−デヒドロゲナーゼ、L−キシルロースリダクターゼ、およびキシトールデヒドロゲナーゼの任意の組合せが挙げられる。これらの酵素は、天然にペントース糖を代謝させる任意の生物に由来し得る。そのような生物の例としては、例えば、Kluyveromyces sp.、Zymomonas sp.、E. coli、Clostridium sp.、およびPichia sp.が挙げられる。
実施例12〜15には、宿主細胞におけるペントース利用経路を促進させるか、またはより効率化させる方法が説明されている。宿主細胞の株の背景は、そのペントース利用経路の効率に影響し得る。宿主細胞がSaccharomyces sp.である本発明の実施形態において、好ましいペントース利用経路は、DA24−16(実施例13参照)およびL2612(実施例16参照)を含んでいる。ペントース利用経路に関与する酵素をコードしているポリヌクレオチドを含んでいる他の宿主細胞としては、DuPont Zymomonas 株(WO 2009/058927)およびSaccharomyces株(米国特許第5,789,210号)が挙げられる。
本発明の一部の実施形態において、宿主細胞は、ペントーストランスポータをコードしている組換えポリヌクレオチドを含んでいる。一部の実施形態において、ペントーストランスポータとしては、本明細書において見いだされ、かつ記載されているトランスポータ(NCU00821、NCU04963、NCU06138、STL12/XUT6、SUT2、SUT3、XUT1、およびXUT3(実施例11参照)が挙げられる)が挙げられる。他の実施形態において、ペントーストランスポータとしては、C. intermedia由来のGxs1、P. stipitis由来のAut1、D. hansenii由来のXylhp(Nobre et al., 1999)、K. marxianus由来のキシローストランスポータ(Stambuk et al., 2003)、Ambrosiozyma monospora由来のLAT1およびLAT2(それぞれEMBL AY923868およびAY923869、R. Verho et al.)、C. arabinofermentans由来のART1(Fonseca et al., 2007)、K. marxiamus由来のKmLAT1(Knoshaug et al., 2007)、P. guilliermondii由来のPgLAT2(Knoshaug et al., 2007)、ならびにP. stipitis由来のaraT(Boles & Keller, 2008)が挙げられ得る。
〔本発明の宿主細胞の生成方法および培養方法〕
本発明は、セロデキストリンまたはペントースを細胞に輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞に関する。本明細書にさらに記載されているのは、宿主細胞へのセロデキストリンの輸送を促進させる方法、セロデキストリンを含有している培地における細胞の成長を促進させる方法、セルロース由来の糖およびヘミセルロース由来の糖を共発酵させる方法、ならびにセロデキストリンを細胞に輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備することによって、炭化水素または炭化水素の誘導体を製造する方法である。本明細書にさらに記載されているのは、宿主細胞へのセロデキストリンの輸送を促進させる方法、セロデキストリンを含有している培地における細胞の成長を促進させる方法、セロデキストリンを細胞に輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備することによって、炭化水素または炭化水素の誘導体を製造する方法である。
本発明は、セロデキストリンまたはペントースを細胞に輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞に関する。本明細書にさらに記載されているのは、宿主細胞へのセロデキストリンの輸送を促進させる方法、セロデキストリンを含有している培地における細胞の成長を促進させる方法、セルロース由来の糖およびヘミセルロース由来の糖を共発酵させる方法、ならびにセロデキストリンを細胞に輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備することによって、炭化水素または炭化水素の誘導体を製造する方法である。本明細書にさらに記載されているのは、宿主細胞へのセロデキストリンの輸送を促進させる方法、セロデキストリンを含有している培地における細胞の成長を促進させる方法、セロデキストリンを細胞に輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備することによって、炭化水素または炭化水素の誘導体を製造する方法である。
本発明の宿主細胞を製造し、培養する方法は、本発明の組換えポリヌクレオチドを含んでいる発現ベクターの、宿主細胞への導入または移入を包含し得る。発現ベクターを宿主細胞に移入させるそのような方法は、当業者にとって公知である。例えば、発現ベクターを用いてE. coliを形質転換させる1つの方法は、カルシウム沈降を介して発現ベクターが導入される塩化カルシウム処理に関する。また、他の塩(例えば、リン酸カルシウム)が、類似の手法にしたがって用いられ得る。さらに、電気穿孔法(すなわち、核酸配列による細胞の透過性を増大させる電流の使用)は、宿主細胞に形質移入するために用いられ得る。また、核酸配列のマイクロインジェクションは、宿主細胞に形質移入する能力をもたらす。また、他の手段(例えば、脂質複合体、リポソーム、およびデンドリマー)が採用され得る。当業者は、これらの方法または他の方法を用いて所望の配列を宿主細胞に形質移入し得る。
ベクターは、自律的に複製するベクター(すなわち、染色体外の構成要素として存在しており、その複製が染色体の複製と独立しているベクター)(例えば、プラスミド、染色体外因子、小染色体または人工染色体)であり得る。ベクターは、自律的な複製を確保する任意の手段を含み得る。代替的に、ベクターは、宿主細胞に導入されるときにゲノムに組み込まれ、組み込まれたゲノムの染色体と共に複製されるベクターであり得る。さらに、宿主細胞のゲノムに導入される総DNAをともに含んでいる、単一もしくは2つ以上のベクターもしくはプラスミド、またはトランスポゾンが用いられ得る。
ベクターは、形質転換された宿主細胞の容易な選択を可能にする1つ以上の選択可能なマーカーを好ましく含んでいる。選択可能なマーカーは、例えば、殺生物性もしくはウイルスに対する耐性、重金属に対する耐性、および栄養要求性に対する原栄養性などである。細菌細胞の選抜は、遺伝子(例えば、amp、gpt、neo、およびhyg遺伝子)によって付与された抗生物質耐性に基づいてなされ得る。
酵母宿主にとって好適なマーカーは、例えば、ADE2、HIS3、LEU2、LYS2、MET3、TRP1、およびURA3である。糸状菌宿主における使用に好適なマーカーは、amdS(アセタミダーゼ)、argB(オルニチン カルバモイルトランスフェラーゼ)、bar(ホスフィノトリシン アセチルトランスフェラーゼ)、hph(ハイグロマイシン ホスホトランスフェラーゼ)、niaD(硝酸塩 レダクターゼ)、pyrG(オロチジン−5’−リン酸塩デカルボキシラーゼ)、sC(硫酸塩アデニルトランスフェラーゼ)、およびtrpC(アントラニル酸塩シンターゼ)などである。Aspergillusにおける使用にとって好ましいのは、Aspergillus nidulansまたはAspergillus oryzaeのamdS遺伝子およびpyrG遺伝子であり、Streptomyces hygroscopicusのbar遺伝子である。Trichodermaにおける使用にとって好ましいのはbarおよびamidSである。
ベクターは、宿主ゲノムへのベクターの組込み、またはゲノムと非依存的に細胞におけるベクターの自律的な複製を可能にする(複数の)因子を含んでいることが好ましい。
宿主ゲノムへの組込みのために、ベクターは、相同性組換えまたは非相同性によるゲノムへのベクターの組込みのための、遺伝子の配列またはベクターの任意の他のエレメントに依存し得る。代替的に、ベクターは、宿主のゲノムへの相同性組換えによる直接の組込みのためにさらなるヌクレオチドを含んでいる。さらなるヌクレオチド配列は、染色体の正確な位置にベクターを組み込むことを可能にする。正確な位置への組込みの可能性を増大させるために、組込みエレメントは、対応する標的配列に対して高い同一性を有して、相同性組換えの可能性を増大させる十分な数(例えば、100〜10000塩基対、好ましくは400〜10000塩基対、最も好ましくは800〜10000塩基対)の核酸を含んでいる。組込みエレメントは、宿主のゲノムの標的配列と相同性の任意の配列であり得る。さらに、組込みエレメントは、非翻訳領域または翻訳領域のヌクレオチド配列を有しているヌクレオチドの配列であり得る。一方、ベクターは非相同性組換えによって宿主のゲノムに組み込まれ得る。
自律的な複製のために、ベクターは、該当する宿主においてベクターを自律的に複製させることを可能にする複製の開始点をさらに含んでいる。複製の開始点は、細胞において機能する自律的な複製を媒介する任意のプラスミドレプリケータであり得る。“複製の開始点”または“プラスミドレプリケータ”という用語は、インビボにおけるプラスミドまたはベクターの複製を可能にする配列として本明細書において定義付けられる。酵母宿主における使用のための複製の開始点の例は、複製の2ミクロン開始点、ARS1、ARS4、ARS1およびCEN3の組み合わせ、ならびにARS4およびCEN6の組合せである。糸状菌細胞に有用な複製の開始点の例は、AMA1およびANS1(Gems et al., 1991; Cullen et al., 1987;国際公開第00/24883号)である。AMA1遺伝子の単離、および当該遺伝子を含んでいるプラスミドもしくはベクターの構築は、国際公開第00/24883号に開示されている方法によってなされ得る。
他の宿主にとって、形質転換の手法は、例えば、KluyveromycesについてJeremiah D. Read, et al., Applied and Environmental Microbiology, Aug. 2007, p. 5088-5096、ZymomonasについてOsvaldo Delgado, et al., FEMS Microbiology Letters 132, 1995, 23-26、Pichia stipitisについて米国特許第7,501,275号、およびClostridiumについて国際公開第2008/040387号に見出され得る。
2コピー以上の遺伝子は、遺伝子産物の生産を促進するために宿主に挿入されて、遺伝子産物の産生を増強させ得る。遺伝子のコピー数の増加は、宿主のゲノム内への少なくとも1コピーのさらなる遺伝子の組込みによって実現され得る。代替的に、遺伝子のコピー数の増加は、増幅されたコピー数の選択的なマーカー遺伝子を含んでいる細胞に、ヌクレオチド配列とともに増幅可能な選択的なマーカー遺伝子を含めるて、これによって遺伝子のさらなるコピーが、適切な選択試薬の存在下における細胞の培養によって選択され得ることによって、実現され得る。
本発明の組換え発現ベクターの構築のために上述のエレメントを連結させるために使用される方法は、当業者にとって公知である(例えば、上述のSambrook et al., 1989参照)。
宿主細胞は、少なくとも1つの発現ベクターを用いて形質転換される。1つの発現ベクターのみが用いられるとき(媒介物の添加なしに)、ベクターは、必要な核酸配列のすべてを含んでいる。
いったん宿主細胞が発現ベクターを用いて形質転換されると、宿主細胞は成長可能になる。本発明の方法は、細胞において組換え核酸が発現されている宿主細胞を培養することを包含している。微生物宿主にとって、この過程は好適な培地における細胞の培養を必然的にともなう。細胞は、35℃、適切な培地において典型的に成長させられる。本発明における好ましい成長培地としては、通常の市販の調製培地(例えば、Luria Bertani(LB)培地、Sabouraudデキストロース(SD)培地または酵母培地(YM)培地)が挙げられる。また、組成の明らかな他の成長培地または他の合成の成長培地が使用され得、特定の宿主細胞の成長にとって適切な培地は、微生物学または発酵科学の当業者にとって公知である。温度の幅および他の成長に適切な条件は当該分野において公知である(例えばBailey and Ollis 1986参照)。
本発明のいくつかの局面によれば、培養培地は、宿主細胞のための炭素源を含んでいる。そのような“炭素源”は、原核細胞または単純な真核細胞の成長のための炭素源としての使用に適切な基質または化合物を指す。炭素源は、種々の形態(重合体、炭化水素、酸、アルコール、アルデヒド、ケトン、アミノ酸、ペプチドなどが挙げられるが、これらに限定されない)であり得る。これらのとしては、例えば、単糖(例えば、グルコース)、オリゴ糖、多糖、バイオマス重合体(例えばセルロースまたはヘミセルロース)、キシロース、アラビノース、二糖(例えばスクロース)、飽和脂肪酸、不飽和脂肪酸、コハク酸塩、乳酸塩、酢酸塩、エタノールなど、またはこれらの混合物が挙げられる。炭素源は、さらに光合成の産物(グルコースが挙げられるが、こらに限定されない)であり得る。
好ましい実施形態において、炭素源はバイオマス重合体(例えばセルロースまたはヘミセルロース)である。本明細書に説明されるとき、“バイオマス重合体”は、生物学的な材料に含まれている任意の重合体である。生物学的な物質は、生存しているか、または死亡している。バイオマス重合体としては、例えばセルロース、キシラン、キシロース、ヘミセルロース、リグニン、マンナン、およびバイオマスに通常に見られる他の物質が挙げられる。非限定的なバイオマス重合体の原料の例としては、草本(例えばスイッチグラス、Miscanthus)もみ殻、バガス、綿、ジュート、麻、アマ、竹、サイザル麻、マニラ麻、麦、葉、刈り取った芝、トウモロコシの茎と葉、トウモロコシの穂軸、穀類の蒸留粕、マメ科の植物、ソルガム、サトウキビ、サトウダイコン繊維、材木の切り屑、おが屑、およびバイオマス収穫物(例えば、Crambe)が挙げられる。
適切な炭素源に加えて、培地は、好適なミネラル、塩、補助因子、緩衝液および他の構成要素を含んでいる必要がある。当該他の構成要素は、種々の糖の発酵ならびに炭化水素および炭化水素の誘導体の製造にとって必要な酵素的な経路の促進、ならびに培養物の成長にとって好適な、当業者にとって公知の構成要素である。反応は、好気性条件または嫌気性条件のもとに実施され得、ここで、好気性、無酸素性または嫌気性の条件は、微生物の必要性に基づいて選択される。宿主細胞の成長および/または増殖にしたがって、種々の糖もしくはバイオマス重合体に基づく成長、糖発酵、または炭化水素もしくは炭化水素の誘導体の合成に必要な酵素、トランスポータ、または他のタンパク質の発現は、作用を受ける。
〔細胞への糖の輸送を促進させる方法〕
本発明は細胞への糖の輸送を促進させる方法を提供する。一局面において、本発明は、宿主細胞を準備する第1のステップを包含している、細胞への糖の輸送を促進させる方法を提供する。当該宿主細胞は組換えポリヌクレオチドを含んでいる。当該組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしている。上記第1のステップを包含している上記方法は、以下の1つ以上:膜貫通型のα−ヘリックス1は配列番号1を含んでいる;膜貫通型のα−ヘリックス2は配列番号2を含んでいる;膜貫通型のα−ヘリックス2および膜貫通型のα−ヘリックス3を接続しているループは配列番号3を含んでいる;貫通型のα−ヘリックス3は配列番号3を含んでいる;膜貫通型のα−ヘリックス5は配列番号4を含んでいる;膜貫通型のα−ヘリックス6は配列番号5を含んでいる;膜貫通型のα−ヘリックス6および膜貫通型のα−ヘリックス7の間にある領域は配列番号6を含んでいる;膜貫通型のα−ヘリックス7は配列番号7を含んでいる;膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびこれらの間にある配列は配列番号8を含んでいる、を満たしている。上記方法は、上記組換えポリヌクレオチドが発現されるように上記宿主細胞を培養する第2のステップを包含しており、上記組換えポリヌクレオチドの発現は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、細胞へのセロデキストリンの促進された輸送をもたらす。細胞へのセロデキストリンの輸送は、当業者にとって公知の方法(実施例9に記載の方法が挙げられる)によって測定され得る。実施例9に記載の方法は、例えば細胞への[3H]−セロビオースの取込みを測定する方法またはセロビオースが単一の炭素源であるときのS. cerevisiae宿主細胞の成長能を測定する方法である。上記組換えポリヌクレオチドを含んでいる上記宿主細胞および上記組換えポリヌクレオチドを含んでいない宿主細胞は、それ以外の点では遺伝的背景において典型的に同一である。
本発明は細胞への糖の輸送を促進させる方法を提供する。一局面において、本発明は、宿主細胞を準備する第1のステップを包含している、細胞への糖の輸送を促進させる方法を提供する。当該宿主細胞は組換えポリヌクレオチドを含んでいる。当該組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしている。上記第1のステップを包含している上記方法は、以下の1つ以上:膜貫通型のα−ヘリックス1は配列番号1を含んでいる;膜貫通型のα−ヘリックス2は配列番号2を含んでいる;膜貫通型のα−ヘリックス2および膜貫通型のα−ヘリックス3を接続しているループは配列番号3を含んでいる;貫通型のα−ヘリックス3は配列番号3を含んでいる;膜貫通型のα−ヘリックス5は配列番号4を含んでいる;膜貫通型のα−ヘリックス6は配列番号5を含んでいる;膜貫通型のα−ヘリックス6および膜貫通型のα−ヘリックス7の間にある領域は配列番号6を含んでいる;膜貫通型のα−ヘリックス7は配列番号7を含んでいる;膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびこれらの間にある配列は配列番号8を含んでいる、を満たしている。上記方法は、上記組換えポリヌクレオチドが発現されるように上記宿主細胞を培養する第2のステップを包含しており、上記組換えポリヌクレオチドの発現は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、細胞へのセロデキストリンの促進された輸送をもたらす。細胞へのセロデキストリンの輸送は、当業者にとって公知の方法(実施例9に記載の方法が挙げられる)によって測定され得る。実施例9に記載の方法は、例えば細胞への[3H]−セロビオースの取込みを測定する方法またはセロビオースが単一の炭素源であるときのS. cerevisiae宿主細胞の成長能を測定する方法である。上記組換えポリヌクレオチドを含んでいる上記宿主細胞および上記組換えポリヌクレオチドを含んでいない宿主細胞は、それ以外の点では遺伝的背景において典型的に同一である。
一部の実施形態において、上記ポリペプチドは、少なくとも29%、少なくとも30%、少なくとも35%、少なくとも40%、少なくとも45%、少なくとも50%、少なくとも55%、少なくとも60%、少なくとも65%、少なくとも70%、少なくとも75%、少なくとも80%、少なくとも85%、少なくとも90%、少なくとも95%、少なくとも99%、または少なくとも100%の、NCU00801またはNCU08114に対するアミノ酸同一性を有している。一部の実施形態において、宿主細胞は、β−グルコシダーゼの触媒作用ドメインをコードしている組換えポリヌクレオチドを含んでいる。そのような実施形態は、セロデキストリンの内因性の利用能を欠いている宿主細胞にとって有用である。好ましくは、β−グルコシダーゼの触媒作用ドメインは細胞内存性であることが好ましい。好ましい実施形態において、β−グルコシダーゼはNeurospora crassa由来である。特に好ましい実施形態において、β−グルコシダーゼはNCU00130によってコードされている。
細胞へのセロデキストリンの輸送を促進させる方法において、上記細胞は、改変された細胞から得られた、セルラーゼ含有の酵素混合物を含んでいる培地において培養され得、セルラーゼ含有の当該酵素混合物は、改変されていない生物から得られた、セルラーゼ含有の酵素混合物と比べて、低いセルラーゼ活性を有している。上記生物は、例えばβ−グルコシダーゼをコードしている遺伝子の変異または標的化されたRNA干渉などによって、β−グルコシダーゼ発現を低下させるために改変され得る。
他の局面において、本発明は、NCU00821またはSTL12/XUT6であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備するステップ;当該組換えポリヌクレオチドが発現されるように当該宿主細胞を培養するステップを包含している、細胞へのキシロースの輸送を促進させる方法を提供する。上記方法において、上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて細胞へのキシロースの促進された輸送をもたらす。
他の局面において、本発明は、XUT1であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備するステップ;当該組換えポリヌクレオチドが発現されるように当該宿主細胞を培養するステップを包含している、細胞へのアラビノースの輸送を促進させる方法を提供する。上記方法において、上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて細胞へのアラビノースの促進された輸送をもたらす。
さらなる他の局面において、本発明は、NCU06138であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備するステップ;当該組換えポリヌクレオチドが発現されるように当該宿主細胞を培養するステップを包含している、細胞へのグルコースの輸送を促進させる方法を提供する。上記方法において、上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて細胞へのグルコースの促進された輸送をもたらす。
さらなる他の局面において、本発明は、SUT2、SUT3またはXUT3であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備するステップ;当該組換えポリヌクレオチドが発現されるように当該宿主細胞を培養するステップを包含している、細胞へのキシロースまたはグルコースの輸送を促進させる方法を提供する。上記方法において、上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて細胞へのキシロースまたはグルコースの促進された輸送をもたらす。他の局面において、本発明は、NCU04963ポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備するステップ;当該組換えポリヌクレオチドが発現されるように当該宿主細胞を培養するステップを包含している、細胞へのキシロース、アラビノースまたはグルコースの輸送を促進させる方法を提供する。上記方法において、上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて細胞へのキシロース、アラビノースまたはグルコースの促進された輸送をもたらす。
他の局面において、本発明は、XUT1であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備するステップ;当該組換えポリヌクレオチドが発現されるように当該宿主細胞を培養するステップを包含している、細胞へのアラビノースの輸送を促進させる方法を提供する。上記方法において、上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて細胞へのアラビノースの促進された輸送をもたらす。
さらなる他の局面において、本発明は、NCU06138であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備するステップ;当該組換えポリヌクレオチドが発現されるように当該宿主細胞を培養するステップを包含している、細胞へのグルコースの輸送を促進させる方法を提供する。上記方法において、上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて細胞へのグルコースの促進された輸送をもたらす。
さらなる他の局面において、本発明は、SUT2、SUT3またはXUT3であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備するステップ;当該組換えポリヌクレオチドが発現されるように当該宿主細胞を培養するステップを包含している、細胞へのキシロースまたはグルコースの輸送を促進させる方法を提供する。上記方法において、上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて細胞へのキシロースまたはグルコースの促進された輸送をもたらす。他の局面において、本発明は、NCU04963ポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備するステップ;当該組換えポリヌクレオチドが発現されるように当該宿主細胞を培養するステップを包含している、細胞へのキシロース、アラビノースまたはグルコースの輸送を促進させる方法を提供する。上記方法において、上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて細胞へのキシロース、アラビノースまたはグルコースの促進された輸送をもたらす。
細胞へのキシロース、アラビノースまたはグルコースの輸送は、当該分野に公知の任意の方法(実施例11に記載されている方法が挙げられる)によって測定され得る。これらの方法としては、例えば、蓄積したD−キシロースとキシリトールと、またはL−アラビノースとアラビニトールとを、上記細胞から浸透圧を用いて抽出し、高速液体クロマトグラフィーを用いてそれを分析することによってD−キシロースまたはL−アラビノースの輸送を測定すること、ならびに単独の炭素源としてのグルコースに基づく成長能を欠いている宿主細胞細胞を用いてグルコースの輸送を測定することが挙げられる。
また、一部の実施形態において、上記宿主細胞は、ペントース利用に関与する1つ以上の酵素をコードしている1つ以上の組換えポリヌクレオチドを含んでいる。1つ以上の上記酵素は、例えば、L−アラビノースイソメラーゼ、L−リブロキナーゼ、L−リブロース−5−P 4エピメラーゼ、キシロースイソメラーゼ、キシルロキナーゼ、アルドースリダクターゼ、L−アラビニトール 4−デヒドロゲナーゼ、L−キシルロースリダクターゼ、およびキシトールデヒドロゲナーゼ、または当該分野に公知の任意の他のペントース利用酵素であり得る。
〔細胞の成長を促進させる方法〕
本発明は、細胞の成長を促進させる方法をさらに提供する。一局面において、本発明は、宿主細胞を準備する第1のステップを包含している細胞の成長を促進させる方法を提供する。上記宿主細胞は、ポリペプチドをコードしている組換えポリヌクレオチドを含んでおり、当該ポリペプチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびα−ヘリックス12を含んでおり、ここで、以下の1つ以上:膜貫通型のα−ヘリックス1は配列番号1を含んでいる、膜貫通型のα−ヘリックス2は配列番号2を含んでいる、膜貫通型のα−ヘリックス2および膜貫通型のα−ヘリックス3を接続しているループは配列番号3を含んでいる、膜貫通型のα−ヘリックス5は配列番号4を含んでいる、膜貫通型のα−ヘリックス6および膜貫通型のα−ヘリックス7の間にある配列は配列番号6を含んでいる、膜貫通型のα−ヘリックス7は配列番号7を含んでいる、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびこれらの間にある配列は配列番号8を含んでいる、ならびに上記ポリペプチドはセロデキストリンを輸送するが、満たされている。上記方法は、セロデキストリンを含んでいる培地において上記宿主細胞を培養する第2のステップを包含しており、ここで、上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。宿主細胞細胞の成長速度は当業者にとって公知の任意の方法によって測定され得る。典型的に、細胞の成長速度は、懸濁物における細胞の濃度の、吸光度による評価によって測定される。上記組換えポリヌクレオチドを含んでいる上記宿主細胞および上記組換えポリヌクレオチドを含んでいない宿主細胞は、それ以外の点では遺伝的背景において同一であることが好ましい。セロデキストリンを含んでいる培地はバイオマス重合体(例えばセルロース)の酵素処理に基づいて生じ得る。
本発明は、細胞の成長を促進させる方法をさらに提供する。一局面において、本発明は、宿主細胞を準備する第1のステップを包含している細胞の成長を促進させる方法を提供する。上記宿主細胞は、ポリペプチドをコードしている組換えポリヌクレオチドを含んでおり、当該ポリペプチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびα−ヘリックス12を含んでおり、ここで、以下の1つ以上:膜貫通型のα−ヘリックス1は配列番号1を含んでいる、膜貫通型のα−ヘリックス2は配列番号2を含んでいる、膜貫通型のα−ヘリックス2および膜貫通型のα−ヘリックス3を接続しているループは配列番号3を含んでいる、膜貫通型のα−ヘリックス5は配列番号4を含んでいる、膜貫通型のα−ヘリックス6および膜貫通型のα−ヘリックス7の間にある配列は配列番号6を含んでいる、膜貫通型のα−ヘリックス7は配列番号7を含んでいる、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびこれらの間にある配列は配列番号8を含んでいる、ならびに上記ポリペプチドはセロデキストリンを輸送するが、満たされている。上記方法は、セロデキストリンを含んでいる培地において上記宿主細胞を培養する第2のステップを包含しており、ここで、上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。宿主細胞細胞の成長速度は当業者にとって公知の任意の方法によって測定され得る。典型的に、細胞の成長速度は、懸濁物における細胞の濃度の、吸光度による評価によって測定される。上記組換えポリヌクレオチドを含んでいる上記宿主細胞および上記組換えポリヌクレオチドを含んでいない宿主細胞は、それ以外の点では遺伝的背景において同一であることが好ましい。セロデキストリンを含んでいる培地はバイオマス重合体(例えばセルロース)の酵素処理に基づいて生じ得る。
一部の実施形態において、上記ポリペプチドは、少なくとも29%、少なくとも30%、少なくとも35%、少なくとも40%、少なくとも45%、少なくとも50%、少なくとも55%、少なくとも60%、少なくとも65%、少なくとも70%、少なくとも75%、少なくとも80%、少なくとも85%、少なくとも90%、少なくとも95%、少なくとも99%または少なくとも100%の、NCU00801またはNCU08114に対するアミノ酸同一性を有している。また、一部の実施形態において、上記宿主細胞は、β−グルコシダーゼの触媒作用ドメインをコードしている組換えポリヌクレオチドを含んでいる。当該実施形態は、セロデキストリンの利用能を欠いている宿主細胞にとって有用である。β−グルコシダーゼの触媒作用ドメインは細胞内存性であることが好ましい。好ましい実施形態において、上記β−グルコシダーゼはNeurospora crassaに由来する。特に好ましい実施形態において、上記β−グルコシダーゼはNCU00130によってコードされている。
細胞の成長を促進させる方法において、培養培地は、改変された生物から得られた、セルラーゼ含有の酵素混合物を含み得、当該混合物は、改変されていない生物から得られた、セルラーゼ含有の酵素混合物と比べて、低いセルラーゼ活性を有している。上記生物は、例えばβ−グルコシダーゼをコードしている遺伝子の変異または標的化されたRNA干渉などによって、β−グルコシダーゼ発現を低下させるために改変され得る。
他の局面において、本発明は、宿主細胞を準備するステップ、およびキシロースを含んでいる培地において当該宿主細胞を培養するステップを包含している、細胞の成長を促進させる方法を提供する。当該宿主細胞は、NCU00821またはSTL12/XUT6であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる。当該ポリペプチドはキシロースを輸送し、上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。他の局面において、本発明は、宿主細胞を準備するステップ、およびアラビノースを含んでいる培地において当該宿主細胞を培養するステップを包含している、細胞の成長を促進させる方法を提供する。当該宿主細胞は、XUT1であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる。当該ポリペプチドはアラビノースを輸送し、上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。他の局面において、本発明は、宿主細胞を準備するステップ、ならびにアラビノースおよびグルコースを含んでいる培地において当該宿主細胞を培養するステップを包含している、細胞の成長を促進させる方法を提供する。当該宿主細胞は、NCU06138であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる。当該ポリペプチドはアラビノースおよびグルコースを輸送し、上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。他の局面において、本発明は、宿主細胞を準備するステップ、ならびにキシロースまたはグルコースを含んでいる培地において当該宿主細胞を培養するステップを包含している、細胞の成長を促進させる方法を提供する。当該宿主細胞は、SUT2、SUT3またはXUT3であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる。当該ポリペプチドはキシロースまたはグルコースを輸送し、上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。他の局面において、本発明は、宿主細胞を準備するステップ、ならびにキシロース、アラビノースおよびグルコースを含んでいる培地において当該宿主細胞を培養するステップを包含している、細胞の成長を促進させる方法を提供する。当該宿主細胞は、NCU04963であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる。当該ポリペプチドは、キシロース、アラビノースおよびグルコースを輸送し、上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。
宿主細胞の成長速度は当業者にとって公知の任意の方法によって測定され得る。典型的に、細胞の成長速度は、懸濁物における細胞の濃度の、吸光度による評価によって測定される。上記組換えポリヌクレオチドを含んでいる上記宿主細胞および上記組換えポリヌクレオチドを含んでいない宿主細胞は、それ以外の点では遺伝的背景において同一であることが好ましい。キシロースまたはアラビノースを含んでいる培地は、バイオマス重合体(例えばヘミセルロース)の酵素処理に基づいて生じ得る。グルコースを含んでいる培地はバイオマス重合体(例えばセルロース)の酵素処理に基づいて生じ得る。
また、一部の実施形態において、上記宿主細胞は、ペントース利用に関与する1つ以上の酵素をコードしている1つ以上の組換えポリヌクレオチドを含んでいる。1つ以上の上記酵素は、例えばL−アラビノースイソメラーゼ、L−リブロキナーゼ、L−リブロース−5−P 4 エピメラーゼ、キシロースイソメラーゼ、キシルロキナーゼ、アルドースリダクターゼ、L−アラビニトール 4−デヒドロゲナーゼ、L−キシルロースリダクターゼ、キシトールデヒドロゲナーゼ、または当該分野に公知の他のペントース利用酵素であり得る。
一局面において、本発明は、バイオマス重合体に基づく細胞の成長を促進させる方法を提供する。好ましい実施形態において、上記バイオマス重合体はセルロースである。好ましい他の実施形態において、上記バイオマス重合体はヘミセルロースである。本発明の一局面によれば、上記方法は、NCU07705であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備することを包含している。本発明の他の局面によれば、上記方法は、上記バイオマス重合体を含んでいる培地において上記宿主細胞を培養することを包含しており、当該宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。
本発明の他の局面において、本発明は、宿主細胞を準備するステップ、およびヘミセルロースを含んでいる培地において当該宿主細胞細胞を培養するステップを包含している、細胞の成長を促進させる方法を提供する。ここで、上記宿主細胞は、NCU01517、NCU09133またはNCU10040であるポリペプチドをコードしている組換えポリヌクレオチドを含んでおり、上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。
本発明の他の局面によれば、上記方法は、NCU05137であるポリペプチドをコードしている外因性のポリヌクレオチドを含んでいる宿主細胞を準備することを包含している。本発明の他の局面によれば、上記方法は、上記外因性のポリヌクレオチドの発現を抑制すること、およびバイオマス重合体を含んでいる培地において上記宿主細胞を培養することを包含しており、ここで、上記宿主細胞は、上記外因性のポリヌクレオチドが抑制されていない細胞と比べて、上記培地においてより早く成長する。
本発明の方法は、上記宿主細胞において組換え体の核酸が発現されるように当該宿主細胞を培養することを包含し得る。微生物の宿主にとって、この過程は好適な培地における細胞の培養を必然的にともなう。典型的に、細胞は、好適な培地、35℃において成長させられる。本発明において好ましい成長培地としては、例えば市販されている通常の好ましい培地(例えばLuria Bertani(LB)ブロス、Sabouraud Dextrose(SD)ブロスまたはYeast medium(YM)ブロス)が挙げられる。また、特徴付けられている他の成長培地または合成の他の成長培地が使用され得、特定の宿主細胞の成長にとって適切な培地は微生物学または発酵科学の分野における当業者によって知られている。成長にとって好適な温度範囲および他の条件は当該分野において公知である(例えば、Bailey and Ollis 1986を参照すればよい)。
上記培地における上記バイオマス重合体の供給源としては、例えば、草本(例えばスイッチグラス、Miscanthus)、もみ殻、バガス、綿、ジュート、麻、アマ、サイザル繊維、タケ、麦わら、葉、刈り取った芝、トウモロコシの茎および葉、トウモロコシの穂軸、穀類の蒸留粕、マメ科の植物、サトウモロコシ、サトウキビ、テンサイの果肉、材木の切り屑、おが屑、ならびにバイオマス収穫物(例えばCrambe)が挙げられ得る。バイオマス重合体に加えて、上記培地は、培養物の成長にとって好適な、ミネラル、補助因子、緩衝液、および当業者にとって公知の他の構成要素を含んでいる必要がある。上記宿主細胞の成長速度は当業者にとって公知の任意の方法によって測定され得る。
本発明の一部の実施形態において、セルラーゼの発現は、組換えポリヌクレオチドの発現によって上記宿主細胞細胞において向上されている。本明細書に使用されるとき“セルラーゼ”は、より短いセロ−オリゴ糖のオリゴマー、セロビオースおよび/またはグルコースに、セルロース重合体を加水分解できる酵素のカテゴリを指す。セルラーゼとしては、限定されることなく、エキソグルカナーゼ、エキソセロビオヒドロラーゼ、エンドグルカナーゼおよびグルコシダーゼが挙げられる。
本発明の一部の実施形態において、ヘミセルラーゼの発現は、組換えポリヌクレオチドの発現によって上記宿主細胞において向上している。本明細書に使用されるとき“ヘミセルラーゼ”は、ヘミセルロース重合体を加水分解できる酵素のカテゴリを指す。ヘミセルラーゼとしては、限定されることなく、キシラナーゼ、マンナーゼ、アラビナーゼ(エンドおよび/またはエキソの種の両方)、およびそれらの対応するグルコシダーゼキシラナーゼ、マンナーゼ、アラビナーゼ(エンドおよび/またはエキソの種の両方)、およびそれらの対応するグルコシダーゼが挙げられる。ヘミセルラーゼの発現はRT−PCRまたは当該分野に公知の他の方法によって測定され得る。
上記外因性のポリヌクレオチドの発現の抑制は、例えば遺伝子の発現、当該遺伝子の機能、または遺伝子産物(すなわち当該遺伝子によってコードされているタンパク質)の機能の低下をもたらす遺伝学的な修飾によって、実現され得る。当該遺伝学的な修飾は、遺伝子の(完全または部分的な)不活性化、欠失、中断、阻害、サイレンシング、または下方制御もしくは遺伝子発現の低下と呼ばれ得る。例えば、そのような遺伝子によってコードされているタンパク質の機能の低下をもたらす遺伝子における遺伝学的な修飾は、(1)当該遺伝子の完全な欠失(すなわち当該遺伝子が存在しておらず、したがってタンパク質が存在しない)、(2)当該タンパク質の不完全な翻訳もしくは翻訳の消失をもたらす(例えば当該タンパク質が発現されない)当該遺伝子における変異、または(3)当該タンパク質の本来の機能を低下させるか、もしくは破壊する(例えば、酵素活性または酵素作用の低下しているか、または消失しているタンパク質が発現される)当該遺伝子における変異の結果であり得る。より詳細には、本明細書において述べられているタンパク質の作用を低下させることに対する言及は、当該タンパク質の低下した発現および/または機能性(生物学的活性)をもたらす、該当する上記宿主細胞における任意の遺伝学的な修飾を一般的に指しており、当該タンパク質の低下した活性(例えば低下した輸送)、当該タンパク質の促進された抑制、または当該タンパク質の促進された分解、ならびに当該タンパク質の発現の低下または消失を包含している。例えば、本発明のタンパク質の作用または活性は、当該タンパク質の産生の抑制もしくは減少、当該タンパク質の低下した作用、または当該タンパク質の作用の抑制によって、低下させられ得る。また、これらの修飾のうちいくつかの組合せが考えられる。タンパク質の産生の阻害または低下は、成長培地における誘導性化合物の存在を必要とするプロモータの制御下に、当該タンパク質をコードしている遺伝子を配置することを包含し得る。誘導物質が培地から枯渇させるような条件を確立することによって、上記タンパク質をコードしている上記遺伝子の発現(したがってタンパク質合成)は、停止させられ得る。また、タンパク質の作用の阻害または低下は、米国特許第4,743,546号に記載されているものと類似の切取り技術の手法を用いることを包含している。この手法を用いるために、所定の上記タンパク質をコードしている上記遺伝子は、ゲノムからの当該遺伝子の制御された特異的な切取りを可能にする特定の遺伝子配列の間にクローン化される。切取りは、例えば米国特許第4,743,546号に記載のような培養物の培養温度の変更、またはいくつかの他の物理的シグナルまたは栄養性シグナルによって、刺激され得る。
本発明の一部の実施形態において、上記宿主細胞のセルラーゼ活性は外因性のポリヌクレオチドの発現を抑制することによって向上する。セルラーゼ活性は、実施例5に記載のように測定され得るか、または当該分野に公知の任意の他の方法によって測定され得る。
本発明の一部の実施形態において、上記宿主細胞のヘミセルラーゼ活性は、外因性のポリヌクレオチドの発現を抑制することによって向上される。ヘミセルラーゼ活性は、実施例17に記載のように測定され得るか、または当該分野に公知の任意の他の方法によって測定され得る。
〔共発酵の方法〕
本発明の一局面は、セルロース由来の糖およびヘミセルロース由来の糖を共発酵させる方法を提供する。本明細書に使用されるとき、共発酵は、同じ容器における2以上の糖の、宿主細胞による同時の使用を指す。上記方法は、宿主細胞を準備するステップ、およびセルロース由来の糖およびヘミセルロース由来の糖を含んでいる培地において当該宿主細胞を培養するステップを包含している。ここで、上記宿主細胞は、セロデキストリントランスポータをコードしている第1の組換えポリヌクレオチドおよびβ−グルコシダーゼの触媒作用ドメインをコードしている第2の組換えポリヌクレオチドを含んでいる。上記組換えポリヌクレオチドの発現は、セルロース由来の糖およびヘミセルロース由来の糖の共発酵を可能にする。
本発明の一局面は、セルロース由来の糖およびヘミセルロース由来の糖を共発酵させる方法を提供する。本明細書に使用されるとき、共発酵は、同じ容器における2以上の糖の、宿主細胞による同時の使用を指す。上記方法は、宿主細胞を準備するステップ、およびセルロース由来の糖およびヘミセルロース由来の糖を含んでいる培地において当該宿主細胞を培養するステップを包含している。ここで、上記宿主細胞は、セロデキストリントランスポータをコードしている第1の組換えポリヌクレオチドおよびβ−グルコシダーゼの触媒作用ドメインをコードしている第2の組換えポリヌクレオチドを含んでいる。上記組換えポリヌクレオチドの発現は、セルロース由来の糖およびヘミセルロース由来の糖の共発酵を可能にする。
第1の組換えポリヌクレオチドは、細胞にセロデキストリンを輸送可能な任意のタンパク質をコードし得る。セロデキストリン輸送は当業者にとって公知の任意の方法(実施例9に記載の方法が挙げられる)によって測定され得る。好ましい実施形態において、第1の組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびα−ヘリックス12を含んでいるポリペプチドをコードしており、ここで、以下の1つ以上:膜貫通型のα−ヘリックス1は配列番号1を含んでいる、膜貫通型のα−ヘリックス2は配列番号2を含んでいる、膜貫通型のα−ヘリックス2および膜貫通型のα−ヘリックス3を接続しているループは配列番号3を含んでいる、膜貫通型のα−ヘリックス5は配列番号4を含んでいる、膜貫通型のα−ヘリックス6および膜貫通型のα−ヘリックス7の間にある配列は配列番号6を含んでいる、膜貫通型のα−ヘリックス7は配列番号7を含んでいる、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびこれらの間にある配列は配列番号8を含んでいる、ならびに上記ポリペプチドはセロデキストリンを輸送する、が満たされている。そのようなポリペプチドの例としては、NCU00801、NCU00809、NCU08114、XP_001268541.1およびLAC2が挙げられる。好ましい実施形態において、上記第1の組換えポリヌクレオチドはNCU00801をコードしている。
上記第2のポリヌクレオチドは、グルコースの放出をともなうβ−D−グルコシドにおける末端にある非還元性残基の加水分解を触媒可能な任意の触媒作用ドメインをコードし得る。上記β−グルコシダーゼの触媒作用ドメインは上記宿主細胞の細胞内に存在していることが好ましい。好ましい実施形態において、β−グルコシダーゼの供給源はNCU00130である。異なる供給源からの触媒作用ドメインは、異なるセロデキストリントランスポータをともなって最良に機能する。
また、一部の実施形態において、上記宿主細胞は、ペントース利用に関与する1つ以上の酵素をコードしている1つ以上の組換えポリヌクレオチドを含んでいる。代替的に、ペントース利用に関与する1つ以上の酵素をコードしている1つ以上の組換えポリヌクレオチドは、上記宿主細胞にとって外因性であり得る。1つ以上の上記酵素としては、例えば、L−アラビノースイソメラーゼ、L−リブロキナーゼ、L−リブロース−5−P 4 エピメラーゼ、キシロースイソメラーゼ、キシルロキナーゼ、アルドースリダクターゼ、L−アラビニトール 4−デヒドロゲナーゼ、L−キシルロースリダクターゼ、キシトールデヒドロゲナーゼ、または当業者にとって公知の任意の他のペントース利用酵素が挙げられ得る。
一部の実施形態において、上記宿主細胞はペントーストランスポータをコードしている第3の組換えポリヌクレオチドを含んでいる。代替的に、上記宿主細胞はペントーストランスポータをコードしている外因性のポリヌクレオチドを含み得る。好ましい実施形態において、上記ペントーストランスポータは、上記宿主細胞にキシロースおよび/またはアラビノースを輸送する。一部の実施形態において、上記第3の組換えポリヌクレオチドは、NCU00821、NCU04963、NCU06138、STL12/XUT6、SUT2、SUT3、XUT1またはXUT3といったポリペプチドをコードしている。ペントースとともにグルコースが存在している成長培地である場合に、上記宿主細胞細胞におけるペントーストランスポータの発現は、共発酵の効率を向上させ得る。
本明細書に記載の共発酵の方法において、セルロース由来の糖としては、セロビオース、セロトリオースおよびセロテトラオースが好ましく挙げら、ヘミセルロース由来の糖としては、キシロースおよびアラビノースが好ましく挙げられる。典型的に、宿主細胞による共発酵のための上記セルロース由来の糖およびヘミセルロース由来の糖を調製するために、リグノセルロース性のバイオマスはまず前処理されてその構造を変化させ、セルロースのより良好な酵素的加水分解を可能にする。前処理は、物理的または化学的な方法(例えば、アンモニア繊維/凍結爆発(ammonia fiber/freeze explosion)、水酸化カルシウムもしくは水酸化ナトリウムに基づく石灰法、および酸触媒ありもしくはなしの蒸気爆発が挙げられる)を包含し得る。酸処理は、リグノセルロース性のバイオマスのヘミセルロース成分からキシロースおよびアラビノースを放出させる。次に、前処理されたバイオマスのセルロース成分は、セルラーゼ混合物によって加水分解されることが好ましい。市販のセルラーゼの例としては、Celluclast 1.5L(登録商標)(Novozymes)、Spezyme CP(登録商標)(Genencor)(Scott W. Pryor, 2010, Appl Biochem Biotechnol)、およびCellulyve 50L(Lyven)が挙げられる。
セルラーゼ混合物は、エンドグルカナーゼ、エキソグルカナーゼおよびβ−グルコシダーゼを典型的に含んでいる。本明細書に記載の共発酵の方法において、上記セルラーゼ混合物におけるβ−グルコシダーゼ活性の程度は、可能な限り最小化されるべきである。例えば、培養培地は、改変された生物から得られた、セルラーゼ含有の酵素混合物を含み得る。当該混合物は、改変されていない生物から得られた、セルラーゼ含有の酵素混合物と比べて、低下したβ−グルコシダーゼ活性を有している。上記生物は、例えばβ−グルコシダーゼをコードしている遺伝子の変異または標的化されたRNA干渉などによって、β−グルコシダーゼの発現を低下させるために改変され得る。
実施例17に記載の通り、意外にも本発明の方法によるセルロースおよびキシロースの共発酵は、上記宿主細胞による糖消費およびエタノール産生に対する相乗効果をもたらした。
〔炭化水素または炭化水素の誘導体の合成方法〕
本発明の一局面は、宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法を提供する。
本発明の一局面は、宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法を提供する。
本明細書に使用されるとき“炭化水素”は、全体的に水素および炭素からなる有機化合物である。炭化水素としては、限定されることなく、メタン、エタン、エテン、エチン、プロパン、プロペン、プロピン、シクロプロパン、アレン、ブタン、イソブタン、ブテン、ブチン、シクロブタン、メチルシクロプロパン、ブタジエン、ペンタン、イソペンタン、ネオペンタン、ペンテン、ペンチン、シクロペンタン、メチルシクロブタン、エチルシクロプロパン、ペンタジエン、ヘキサン、ヘキセン、ヘキシン、シクロヘキサン、メチルシクロペンタン、エチルシクロブタン、プロピルシクロプロパン、ヘキサジエン、ヘプタン、ヘプテン、ヘプチン、シクロヘプタン、メチルシクロヘプタン、ヘプタジエン、オクタン、オクテン、オクチン、シクロオクタン、オクタジエン、ノナン、ノネン、ノニン、シクロノナン、ノナジエン、デカン、デセン、デシン、シクロデカンおよびデカジエンが挙げられる。
本明細書に使用されるとき“炭化水素の誘導体”は、炭素、および水素以外である少なくとも1つの他の元素の有機化合物である。炭化水素の誘導体としては、限定されることなく、アルコール(例えば、アラビニトール、ブタノール、エタノール、グリセロール、メタノール、1,3−プロパンジオール、ソルビトールおよびキシリトール);有機酸(例えば、酢酸、アジピン酸、アスコルビン酸、クエン酸、2,5−ジケト−D−グルコン酸、蟻酸、フマル酸、グルカル酸、グルコン酸、グルクロン酸、グルタル酸、3−ヒドロプロピオン酸、イタコン酸、乳酸、リンゴ酸、マロン酸、プロピオン酸、コハク酸およびキシロン酸);エステル;ケトン(例えば、アセトン);アルデヒド(例えば、フルフラール);アミノ酸(例えば、アスパラギン酸、グルタミン酸、グリシン、リジン、セリンおよびスレオニン);気体(例えば、二酸化炭素および一酸化炭素)が挙げられる。
好ましい実施形態において、上記炭化水素または炭化水素の誘導体は燃料として使用され得る。特に好ましい実施形態において、上記炭化水素または炭化水素の誘導体はエタノールまたはブタノールである。
本発明の一局面によれば、宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法は、宿主細胞を準備する第1のステップを包含している。当該宿主細胞は、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびα−ヘリックス12を含んでいるポリペプチドをコードしているポリヌクレオチドを含んでおり、ここで、以下の1つ以上:膜貫通型のα−ヘリックス1は配列番号1を含んでいる、膜貫通型のα−ヘリックス2は配列番号2を含んでいる、膜貫通型のα−ヘリックス2および膜貫通型のα−ヘリックス3を接続しているループは配列番号3を含んでいる、膜貫通型のα−ヘリックス5は配列番号4を含んでいる、膜貫通型のα−ヘリックス6および膜貫通型のα−ヘリックス7の間にある配列は配列番号6を含んでいる、膜貫通型のα−ヘリックス7は配列番号7を含んでいる、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびこれらの間にある配列は配列番号8を含んでいる、が満たされている。上記ポリペプチドは炭化水素または炭化水素の誘導体の合成のためのセロデキストリンを上記宿主細胞に輸送する。上記方法は、上記宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、セロデキストリンまたはセロデキストリン源を含んでいる培地において当該宿主細胞を培養する第2のステップを包含している。ここで、上記宿主細胞へのセロデキストリンの輸送は、上記組換えポリヌクレオチドの発現によって促進される。一部の実施形態において、上記ポリペプチドは、少なくとも29%、少なくとも30%、少なくとも35%、少なくとも40%、少なくとも45%、少なくとも50%、少なくとも55%、少なくとも60%、少なくとも65%、少なくとも70%、少なくとも75%、少なくとも80%、少なくとも85%、少なくとも90%、少なくとも95%、少なくとも99%または少なくとも100%の、NCU00801またはNCU08114に対するアミノ酸同一性を有している。また、一部の実施形態において、上記宿主細胞はっβ−グルコシダーゼの触媒作用ドメインをコードしている組換えポリヌクレオチドを含んでいる。そのような実施形態は、内因性のセロデキストリンの利用能を欠いている宿主細胞にとって有用である。好ましい実施形態において、上記β−グルコシダーゼはNeurospora crassaに由来する。特に好ましい実施形態において、上記β−グルコシダーゼはNCU00130によってコードされている。上記宿主細胞へのセロデキストリンの輸送は、当業者にとって公知の任意の方法(実施例9に記載の方法が挙げられる)によって測定され得る。典型的に、上記セロデキストリン源はセルロースである。
培養培地は、改変された生物から得られた、セルラーゼ含有の酵素混合物を含み得る。当該混合物は、改変されていない生物から得られた、セルラーゼ含有の酵素混合物と比べて、低下したβ−グルコシダーゼ活性を有している。上記生物は、例えばβ−グルコシダーゼをコードしている遺伝子の変異または標的化されたRNA干渉などによって、β−グルコシダーゼの発現を低下させるために改変され得る。
本発明の他の局面によれば、宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法は、宿主細胞を準備するステップ、ならびに当該宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、キシロースまたはキシロース源を含んでいる培地において上記宿主細胞を培養するステップを包含している。上記宿主細胞は、NCU00821またはSTL12/XUT6であるポリペプチドをコードしているポリヌクレオチドを含んでいる。上記ポリペプチドは炭化水素または炭化水素の誘導体の合成のために上記宿主細胞にキシロースを輸送する。上記宿主細胞細胞へのキシロースの輸送は、上記組換えポリヌクレオチドの発現によって促進されている。
他の局面によれば、宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法は、宿主細胞を準備するステップ、ならびに当該宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、アラビノースまたはアラビノース源を含んでいる培地において上記宿主細胞を培養するステップを包含している。上記宿主細胞は、XUT1であるポリペプチドをコードしているポリヌクレオチドを含んでいる。上記ポリペプチドは炭化水素または炭化水素の誘導体の合成のためのアラビノースを上記宿主細胞に輸送する。上記宿主細胞細胞へのアラビノースの輸送は、上記組換えポリヌクレオチドの発現によって促進されている。
他の局面によれば、宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法は、宿主細胞を準備するステップ、ならびに当該宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、アラビノースもしくはグルコースの源、アラビノースまたはグルコースを含んでいる培地において上記宿主細胞を培養するステップを包含している。上記宿主細胞は、NCU06138であるポリペプチドをコードしているポリヌクレオチドを含んでいる。上記ポリペプチドは炭化水素または炭化水素の誘導体の合成のためのアラビノースまたはグルコースを上記宿主細胞にを輸送する。上記宿主細胞細胞へのアラビノースまたはグルコースの輸送は、上記組換えポリヌクレオチドの発現によって促進されている。
他の局面によれば、宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法は、宿主細胞を準備するステップ、ならびに当該宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、キシロースもしくはグルコースの源、キシロースまたはグルコースを含んでいる培地において上記宿主細胞を培養するステップを包含している。上記宿主細胞は、SUT2、SUT3またはXUT3であるポリペプチドをコードしているポリヌクレオチドを含んでいる。上記ポリペプチドは炭化水素または炭化水素の誘導体の合成のためのキシロースまたはグルコースを上記宿主細胞に輸送する。上記宿主細胞細胞へのキシロースまたはグルコースの輸送は、上記組換えポリヌクレオチドの発現によって促進されている。
他の局面によれば、宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法は、宿主細胞を準備するステップ、ならびに当該宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、キシロース、アラビノースもしくはグルコースの源、キシロース、アラビノースまたはグルコースを含んでいる培地において上記宿主細胞を培養するステップを包含している。上記宿主細胞は、NCU04963であるポリペプチドをコードしているポリヌクレオチドを含んでいる。上記ポリペプチドは炭化水素または炭化水素の誘導体の合成のためのキシロース、アラビノースまたはグルコースを上記宿主細胞に輸送する。上記宿主細胞細胞へのキシロース、アラビノースまたはグルコースの輸送は、上記組換えポリヌクレオチドの発現によって促進されている。
上記宿主細胞へのキシロース、アラビノースまたはグルコースの輸送は当業者にとって公知の任意の方法(実施例1に記載の方法が挙げられる)によって測定され得る。典型的に、グルコース源はセルロースであり、キシロースおよびアラビノースの源はヘミセルロースである。
〔セルロースを分解させる方法〕
本発明の一局面はセルロースを分解させる方法を提供する。当該方法は、セルロースを含んでいる組成物を準備する第1のステップを包含している。上記セルロースは、植物材料(例えば、スイッチグラス、Miscanthus、もみ殻、バガス、アマ、タケ、サイザル繊維、アバカ、麦わら、葉、刈り取った芝、トウモロコシの茎および葉、トウモロコシの穂軸、穀類の蒸留粕、マメ科の植物、サトウモロコシ、サトウキビ、テンサイの果肉、材木の切り屑、おが屑、ならびにバイオマス収穫物)に由来することが好ましい。
本発明の一局面はセルロースを分解させる方法を提供する。当該方法は、セルロースを含んでいる組成物を準備する第1のステップを包含している。上記セルロースは、植物材料(例えば、スイッチグラス、Miscanthus、もみ殻、バガス、アマ、タケ、サイザル繊維、アバカ、麦わら、葉、刈り取った芝、トウモロコシの茎および葉、トウモロコシの穂軸、穀類の蒸留粕、マメ科の植物、サトウモロコシ、サトウキビ、テンサイの果肉、材木の切り屑、おが屑、ならびにバイオマス収穫物)に由来することが好ましい。
上記方法は、上記組成物を、改変された生物から得られた、セルラーゼ含有の酵素混合物と接触させる第2のステップを包含している。セルラーゼ含有の上記酵素混合物は、改変されていない生物から得られたセルラーゼ含有の酵素混合物と比べて、低下したβ−グルコシダーゼ活性を有している。上記生物は、β−グルコシダーゼをコードしている遺伝子の変異、または例えばRNA干渉といった手法を用いて、β−グルコシダーゼの発現を低下させることによって改変され得る。上記生物は真菌または細菌であり得る。好ましい実施形態において、上記生物は糸状菌(例えばT. reesei)である。
代替的に、上記方法は、上記組成物を、改変されてβ−グルコシダーゼ活性が低下しているセルラーゼ含有の酵素混合物と接触させる第2のステップを包含している。例えば、セルラーゼ含有の上記酵素混合物は、アフィニティークロマトグラフィーによって変化させられ得る。ここで、β−グルコシダーゼ酵素は、クロマトグラフィーの間に捕捉され、したがって上記混合物から除去される。他の例において、セルラーゼ含有の上記酵素混合物は、当該混合物におけるβ−グルコシダーゼ酵素の、阻害剤を用いた不活性化によって変化させられ得る。市販のセルラーゼ混合物の例としては、Celluclast 1.5L(登録商標)(Novozymes)、Spezyme CP(登録商標)(Genencor)(Scott W. Pryor, 2010, Appl Biochem Biotechnol)、およびCellulite 50L(Lyven)が挙げられる。
本発明はそれらの好ましい特定の実施形態と関連付けて説明されているが、以上の記載は、例証することを目的としており、本発明の範囲を限定しないことが理解されるべきである。本発明の範囲内における他の局面、利点、および変更は、本発明が属する分野における当業者にとって明らかである。
説明されている本発明および以下の実施例は、限定ではなく例証を目的とした本願の説明のために与えられている。
〔実施例1:MiscanthusおよびAvicelに基づいて成長させたN. crassaのトランスクリプトーム分析〕
本実施例において、N. crassaのゲノムの発現プロファイルを、MiscanthusおよびAvicelに基づく成長の間に調べた。単独の炭素源である結晶性セルロース(Avicel)を含んでいるVogel's最少培地において培養したN. crassa(FGSC 2489)の成長およびセルラーゼ活性は、T. reesei(QM9414)と類似していた(図3);N. crassaは約4日間のうちにAvicelを完全に分解した。また、N. crassaはすり潰したMiscanthusの茎に基づいて急速に成長し、機能性のセルラーゼおよびセミセルラーゼの分解能を示した。N. crassaにおける植物の細胞壁の分解に関連するトランスクリプトームを決定するために、我々は、全ゲノムマイクロアレイ(Kasuga and Glass 2008; Tian et al., 2007; Kasuga et al., 2005)を用いて、すり潰したMiscanthusの茎に基づくN. crassaの成長の間における遺伝子発現プロファイルを観察した。スクロースに基づく成長の16時間後に回収したRNAを、Miscanthus培地に基づいて成長させたN. crassaから16時間後、40時間後、5日後および10日後に単離したRNAと比較した(図4;Tian et al., 2009のSupplemental Data, Dataset S1の第1頁を参照;またデータはbioinfo.townsend.yale.edu/browse.jsp, Experiment IDs 52 and 53に見られる)。
本実施例において、N. crassaのゲノムの発現プロファイルを、MiscanthusおよびAvicelに基づく成長の間に調べた。単独の炭素源である結晶性セルロース(Avicel)を含んでいるVogel's最少培地において培養したN. crassa(FGSC 2489)の成長およびセルラーゼ活性は、T. reesei(QM9414)と類似していた(図3);N. crassaは約4日間のうちにAvicelを完全に分解した。また、N. crassaはすり潰したMiscanthusの茎に基づいて急速に成長し、機能性のセルラーゼおよびセミセルラーゼの分解能を示した。N. crassaにおける植物の細胞壁の分解に関連するトランスクリプトームを決定するために、我々は、全ゲノムマイクロアレイ(Kasuga and Glass 2008; Tian et al., 2007; Kasuga et al., 2005)を用いて、すり潰したMiscanthusの茎に基づくN. crassaの成長の間における遺伝子発現プロファイルを観察した。スクロースに基づく成長の16時間後に回収したRNAを、Miscanthus培地に基づいて成長させたN. crassaから16時間後、40時間後、5日後および10日後に単離したRNAと比較した(図4;Tian et al., 2009のSupplemental Data, Dataset S1の第1頁を参照;またデータはbioinfo.townsend.yale.edu/browse.jsp, Experiment IDs 52 and 53に見られる)。
合計760のN. crassa遺伝子は、4つのMiscanthusサンプル間の相対的な発現レベルにおいて、スクロースのサンプルと比べて、統計学的に有意な相違を示した(Tian et al., 2009における第3頁の、Supplemental Data, Dataset S1を参照)。階層クラスタリングによって、これらの遺伝子は3つの主なクラスタに分類されることが示された(図4A)。遺伝子の第1のクラスター(C1;300の遺伝子)は炭素源であるスクロースを含んでいる最少培地において最も高い発現レベルを示した。これらの遺伝子の機能的なカテゴリ(FunCat)解析(Ruepp 2004)によって、リボソームタンパク質および一次代謝に関連する他の機能的なカテゴリー(例えば、呼吸、電子伝達およびアミノ酸代謝)が多く含まれていることが示された(Tian et al., 2009における第4頁の、Supplemental Data, Dataset S1を参照)。第2のクラスター(C2)は、Miscanthusに基づく培養におけるより後の時点(40時間後、10日後)において、最も高い発現レベルを示した327の遺伝子を包含している(図4A)。このクラスターには、Miscanthusに基づく培養のすべての時点において高い相対的な発現レベルを示した89の遺伝子があった。さらなる分析のため、これらの89の遺伝子を、16時間後のMiscanthusに基づく培養から、最も高い発現レベルを示す遺伝子のクラスターに加えた(C3クラスター、下記を参照)。残りの238の遺伝子のFunCat解析(Ruepp 2004)は、1つの機能的なカテゴリー(C−化合物および炭水化物代謝)がいくぶん多いことを示した(Tian et al., 2009における第5頁の、Supplemental Data, Dataset S1を参照)。
142の遺伝子の第3のクラスターは、N. crassaのMiscanthusに基づく成長から16時間後に最も高い相対的な発現レベルを示した(C3、図4A)。Miscanthusに基づく培養のすべての時点において相対的に高い発現レベルを示した89の遺伝子に加えて、これらの142の遺伝子(C3+クラスター;合計231の遺伝子)のFunCat解析(Ruepp 2004)によって、炭素代謝の関連タンパク質(予想のセルラーゼおよびヘミセルラーゼが挙げられる)が多く含まれていることが示された(図4C;Tian et al., 2009における第6頁の、Supplemental Data, Dataset S1を参照)。N. crassaゲノムの23の予想されているセルラーゼ遺伝子のうちの18が、N. crassaのMiscanthusに基づく成長の間に、特に16時間後の時点における発現レベルの有意な増加を示した(図5)。5の遺伝子が、200倍を超える発現レベルの上昇を示した(cbh−1(CBH(I);NCU07340、gh6−2(CBH(II)様遺伝子;NCU09680)、gh6−3(NCU07190)および2つのGH61遺伝子(gh61−4;NCU01050およびNCU07898)))。
植物の細胞壁は、セルロース微小繊維、ヘミセルロース、リグニン、ペクチン、クチンおよびタンパク質から構成される複合構造である。それゆえ、我々は、Miscanthusに基づいて成長させたN. crassaの発現プロファイルを、Avicel(結晶性セルロースの純粋な形態)に基づいて成長させたN. crassaの発現プロファイルと比較した(Tian et al., 2009における第2頁の、Supplemental Data, Dataset S1を参照;また、データはbioinfo.townsend.yale.edu/browse.jsp, Experiment IDs 52 and 53に見られる)。187を超える遺伝子がAvicelに基づくN. crassaの成長の間における相対的な発現レベルの有意な増加を示した。これらの遺伝子のうちの114がC3+クラスターにおける231の遺伝子と共通していた(図4B)。共通する114の遺伝子セットのFunCat解析によって、炭素代謝に関連すると予想されている遺伝子が多く含まれていることが明確に示された(Tian et al., PNAS, 2009における第6頁の、Supplemental Data, Dataset S1を参照)。この遺伝子セットの範囲内において、分泌性タンパク質がさらに多く含まれていた(114の遺伝子産物のうち53が分泌されると予想された)。これらの53の遺伝子のうち32は、植物の細胞壁の分解を担うという注釈を有している予想タンパク質(predicted protein)をコードしている。一方、これらの53の遺伝子のうち16は、いずれも未知機能の推定タンパク質(putative protein)または仮定のタンパク質(hypothetical protein)をコードしている。残りの61の遺伝子は、メジャーファシリテータスーパーファミリーの予想されている10のトランスポータ(NCU00801、NCU00988、NCU01231、NCU04963、NCU05519、NCU05853、NCU05897、NCU06138、NCU08114、およびNCU10021)および23の推定タンパク質または仮定のタンパク質を包含する予想されている細胞内タンパク質をコードしている。
Miscanthus特異的なクラスター(図4B)における117の遺伝子のうち、37は、分泌されると予想タンパク質をコードしている。9の予想されているヘミセルラーゼ、またはヘミセルロースの分解に関連する酵素を同定した(NCU00710、NCU04265、NCU04870、NCU05751、NCU05965、NCU09170、NCU09775、NCU09923、およびNCU09976)(Tian et al., 2009の表2)。残り80のMiscanthus特異的な遺伝子は、予想されている細胞内タンパク質(例えば、NCU00891(キシリトール脱水素酵素)およびNCU00643(予想されているアラビニトール脱水素酵素))をコードしている遺伝子(ペントース糖の代謝に関連する遺伝子が挙げられる)、予想されている糖トランスポータ(NCU01132)をコードしている遺伝子、および48の機能未知のタンパク質をコードしている遺伝子である。
〔実施例2:MiscanthusおよびAvicelに基づいて成長させたN. crassaのセクレトーム分析〕
菌類によるリグノセルロース分解は、細胞外において生じ、細胞壁成分の脱重合に関連するタンパク質の分泌を必要とする(Lynd et al., 2002)。トランスクリプトームのプロファイリングデータ(N. crassaをMiscanthusおよびAvicelに基づいて成長させたとき、予想されているセルラーゼ、ヘミセルラーゼ、および他の分泌されるタンパク質をコードしている遺伝子の発現レベルが増加することを示した)を比較するために、我々はショットガンプロテオミクスの手法を用いてN. crassaのセクレトームを分析した(図4B)。7日間が経過したMiscanthusおよびAvicelにおける培養物に由来する上清をトリプシン処理し、タンデムの液体クロマトグラフィー−ナノエレクトロスプレーイオン化質量分析法(MS;実施例5を参照)によって解析した。リン酸膨潤セルロース(PASC)に結合している分泌タンパク質を濃縮し、同様にMSによって解析した。
菌類によるリグノセルロース分解は、細胞外において生じ、細胞壁成分の脱重合に関連するタンパク質の分泌を必要とする(Lynd et al., 2002)。トランスクリプトームのプロファイリングデータ(N. crassaをMiscanthusおよびAvicelに基づいて成長させたとき、予想されているセルラーゼ、ヘミセルラーゼ、および他の分泌されるタンパク質をコードしている遺伝子の発現レベルが増加することを示した)を比較するために、我々はショットガンプロテオミクスの手法を用いてN. crassaのセクレトームを分析した(図4B)。7日間が経過したMiscanthusおよびAvicelにおける培養物に由来する上清をトリプシン処理し、タンデムの液体クロマトグラフィー−ナノエレクトロスプレーイオン化質量分析法(MS;実施例5を参照)によって解析した。リン酸膨潤セルロース(PASC)に結合している分泌タンパク質を濃縮し、同様にMSによって解析した。
合計50のタンパク質を、タンデムMSによって高い信頼性において同定した(表2および3)。Miscanthusによって成長させたN. crassaの培養物において34のタンパク質が検出され、一方Avicelに基づいて成長させた培養物から38のタンパク質が同定された。これらの22のタンパク質のうち21は、コンピュータによる解析に基づいて、分泌されると予想され、19はMiscanthusおよびAvicelに基づいて成長させた培養物の両方において発現レベルの増加を示した(表2)。重複しているデータセットは、N. crassaにおける23の予想されているセルラーゼのうちの8を包含していた(表3)。また、5のヘミセルラーゼ、1の予想されているβ−グルコシダーゼ(gh3−4;NCU04952)、炭水化物に対する活性が予想されている5のタンパク質、および機能未知の2のタンパク質(NCU07143およびNCU05137)があった(表4〜5)。
表2に関して、注釈は、ウェブページbroad.mit.edu/annotation/genome/neurospora/Home.html.における広範な研究機関によってもたらされた。“検出されたサンプル”は、ペプチドが特定のタンパク質に関して検出されたサンプルである。ペプチドを手動の検査によって確認した。少なくとも1つのペプチドが生物学的な繰返しのそれぞれにおいて検出された場合に、タンパク質は存在していると判断した。“TOTAL”はすべての細胞外タンパク質のトリプシンによる分解から検出されたペプチドを指す。“PASC BOUND”は、リン酸膨潤セルロースに結合するタンパク質を濃縮した後に検出されたペプチドを指す。“UNBOUND”は、PASC結合タンパク質の除去後に溶液に残っていたタンパク質を指す。
Avicelに基づく培養物においてのみ確認された同定された16のタンパク質があり、これらのうち14(2の予想されているセルラーゼ(gh61−1;NCU02240およびgh45−1;NCU05121)、1のキシラナーゼ(gh11−1;NCU02855)、1の予想されているプロテアーゼ(NCU04205)、炭水化物に対する活性が予想されている他の3のタンパク質(NCU08909、NCU05974およびgh30−1(NCU04395))、3のNeurospora特異的な機能未知のタンパク質、および4の保存された仮定のタンパク質(セルロース結合ドメインを有している1のタンパク質(NCU09764)が挙げられる)が挙げられる)が分泌されると予想された(表6)。12のタンパク質がMiscanthusにおける培養物の培養ろ過物に特異的であり、これらのうちの7は分泌されると予想された(表3)。5の予想されている細胞内タンパク質のうちの3は、保存された仮定のタンパク質であった。残りの2としては、予想されているグリオキサルオキシダーゼ(NCU09267、N. crassaのMiscanthusにおけるトランスクリプトームから同定された)およびヌクレオシド二リン酸キナーゼ(ndk−1;NCU04202、N. crassaのトランスクリプトームにおいて同定されなかった)が挙げられる。分泌されると予想される7のタンパク質としては、3の予想されているエステラーゼ(NCU04870、NCU05159、およびNCU08785)、2の予想されているキシラナーゼ(GH51;NCU02343およびGH54;NCU09775)、1のβ−キシロシダーゼ(gh3−7;NCU09923)および保存されている仮定のタンパク質(NCU05751)が挙げられる。
多くの植物の細胞壁分解酵素は、基質に対する酵素の結合を補助するセルロース結合モジュール(CBM)を有している(Linder and Teeri 1996)。N. crassaのゲノムにおいて、19の遺伝子によってコードされているタンパク質が、CBM1ドメインを有していると予想されている(Cantarel et al., 2009)。これらの19の遺伝子のうち16は、Miscanthusに基づいて成長させた培養物において相対的な遺伝子発現の増加を示した。
MSによって同定された50のタンパク質のうち11は、CBM1ドメインを有していた。セルロースに結合するタンパク質を濃縮させるためにPASCを用いた(方法に関して実施例4を参照)。9のタンパク質が、Miscanthusに基づいて成長させたN. crassaの培養物の上清に由来するPASCに結合することがMSによって確認され、一方、Avicelにおける上清に由来する8のタンパク質が同定された(7のセルロース結合タンパク質が両方において同定された)(表2、3、8)。これらとしては、NCU00206、予想されているセロビオースデヒドロゲナーゼ;gh5−1(NCU00762),予想されているエンドグルカナーゼ;NCU05955、予想されているGH74kシログルカナーゼ;gh11−2(NCU07225)、予想されているエンドキシラナーゼ;cbh−1(NCU07340);gh61−5(NCU08760)、予想されているエンドグルカナーゼ;およびgh6−2(NCU09680)、予想されているセロビオースヒドロラーゼ2前駆体が挙げられる。
〔実施例3:トランスクリプトーム/セクレトームのデータセットの重複において同定された複数の遺伝子に欠失を含んでいる株における、細胞外タンパク質およびセルラーゼ活性の性質決定〕
MiscanthusおよびAvicelに基づいて成長させた培養物の両方において検出された22の細胞外タンパク質のうち、16をコードしている遺伝子における欠失を含んでいる同核共存体株(homokaryotic strains)は、一般的に利用可能である(Dunlap et al., 2007)。これらの16の欠失株はいずれも、植物の細胞壁またはセルロース分解に対するそれらの影響のために、N. crassaにおいてこれまで性質決定されていない。16の欠失株を、好ましい炭素源としてスクロースまたはAvicelを含んでいる培地の両方おいて成長させた。すべての株がスクロースに基づく野生型の成長特性を示した。Avicelを含んでいる培地において、16の欠失株のバルク成長を、7日にわたって観察した。7日後、培養ろ過液における分泌された総タンパク質、エンドグルカナーゼ活性、β−グルコシダーゼ活性、および総Avicerase(Avicelase)活性を測定し、すべての変異体の起源である野生型株と比較した(図6)。また、分泌タンパク質の相対的な存在量を調べるために、濃縮していない培養上清に対してSDS−PAGEを行った。
MiscanthusおよびAvicelに基づいて成長させた培養物の両方において検出された22の細胞外タンパク質のうち、16をコードしている遺伝子における欠失を含んでいる同核共存体株(homokaryotic strains)は、一般的に利用可能である(Dunlap et al., 2007)。これらの16の欠失株はいずれも、植物の細胞壁またはセルロース分解に対するそれらの影響のために、N. crassaにおいてこれまで性質決定されていない。16の欠失株を、好ましい炭素源としてスクロースまたはAvicelを含んでいる培地の両方おいて成長させた。すべての株がスクロースに基づく野生型の成長特性を示した。Avicelを含んでいる培地において、16の欠失株のバルク成長を、7日にわたって観察した。7日後、培養ろ過液における分泌された総タンパク質、エンドグルカナーゼ活性、β−グルコシダーゼ活性、および総Avicerase(Avicelase)活性を測定し、すべての変異体の起源である野生型株と比較した(図6)。また、分泌タンパク質の相対的な存在量を調べるために、濃縮していない培養上清に対してSDS−PAGEを行った。
2の予想されているエキソグルカナーゼ(cbh−1;NCU07340およびgh6−2;NCU09680)および予想されているβ−グルコシダーゼ(gh3−4;NCU04952)の欠失を含んでいる株について、Avicelに基づく成長は不良であった。cbh−1の変異体は最も重大な影響を受けており、7日後に多量のAvicelが残っていたが、野性株において同時期にすべてのAvicelが分解されていた。16の欠失株のうち10について、分泌タンパク質のSDS−PAGE分析は、単一のバンドが消失した変異した細胞外タンパク質プロファイルを示した。それゆえ、特定のタンパク質のバンドは予想された遺伝子に基づいていた(図6Aの囲み;図7)。これらとしては、NCU00762(gh5−1)、NCU04952(gh3−4)、NCU05057(gh7−1)、NCU05137、NCU05924(gh10−1)、NCU05955、NCU07190(gh6−3)、NCU07326、NCU07340(cbh−1)、およびNCU09680(gh6−2)が挙げられる。
欠失株のほとんどについて、培養上清に分泌された総タンパク質、エンドグルカナーゼ活性、β−グルコシダーゼ活性、およびAvicerase活性は、野生型株と類似していた(図6B、Cおよび表9)。
この傾向からの逸脱していることが、Δgh5−1(NCU00762)、Δgh3−4(NCU04952)、ΔNCU05137、Δcbh−1(NCU07340)、およびΔgh6−2(NCU09680)変異体に認められた。Δgh5−1(NCU00762)、Δgh3−4(NCU04952)、およびΔcbh−1(NCU07340)において、Avicerase、エンドグルカナーゼまたはΔ−グルコシダーゼ活性が、対応する野生型の活性より低かった。特に、Avicerase酵素アッセイにおけるより高いレベルのセロビオースおよびより低いグルコース、ならびにPNPGアーゼ活性によって示されるように、NCU04952の欠失はすべてのβ−グルコシダーゼ活性を培養上清から消失させた(図6B、C)。エンドグルカナーゼ活性が低下したにもかかわらず、Δgh5−1(NCU00762)由来の培養ろ過液は、Avicerase活性において野生型株と比較して顕著な欠損を示さなかった(図6C)。予測された通り、Δcbh−1(NCU07340)における変異体は、成長不良に起因して、エンドグルカナーゼ活性およびAvicerase活性の低下を生じた。また、Δcbh−1変異体について見られたように、NCU09680(CBH(II)様タンパク質(gh6−2)をコードしている)の欠失を含んでいる株は、セロビオースの蓄積の減少を示した(図6C)。
3つの株における変異は分泌タンパク質(特にCBH(I);gh3−4(NCU04952)、gh7−1(NCU05057)および仮定のタンパク質の遺伝子(NCU05137))のレベルの上昇を引き起こした(図6A)。分泌タンパク質のレベルの上昇に加え、ΔNCU05137変異体はエンドグルカナーゼ、β−グルコシダーゼ、およびAvicerase活性の上昇を示した(図6B、C)。NCU05137は、多数の糸状子嚢菌(セルロース分解性の他の真菌)のゲノムにおいて高度に保存されているが、特にT. reeseiにおいて相同物を有していない(図2)。Δgh3−4、Δgh7−1、およびΔNCU05137において見られるCBH(I)のレベルの上昇は、分泌の増加、タンパク質の安定性、またはそうでなければcbh−1の発現における上昇を引き起こすフィードバックのいずれか一方に起因すると可能性がある。これらの可能性を明確にするために、ΔNCU05137およびΔgh3−4(NCU04952)によって産生される細胞外タンパク質のプロファイルを、定量RT−PCRによって分析して、cbh−1(NCU07340)およびgh6−2(CBH(II);NCU09680)の遺伝子発現レベルと比較した(図8)。Avicelに基づいて成長させた培養物において、早くも2日後に、ΔNCU05137およびΔgh3−4の株は、CBH(I)タンパク質のレベルがより高くなった。Avicelに基づいて成長させた培養物由来のcbh−1およびgh6の定量RT−PCRによって、両方の遺伝子について野生型、ΔNCU05137変異体およびΔgh3−4変異体は、成長から2日後に高い発現レベルを示すことがわかった。しかし、これらの遺伝子の発現は野生株において3日後に著しく減少したが、cbh−1およびgh6−2の発現レベルはΔNCU05137変異体において増加し、Δgh3−4において野生型よりも減少した(図8)。
ΔNCU05137およびΔgh3−4変異体におけるcbh−1およびgh6−2遺伝子の持続発現は、CBH(I)およびCBH(II)タンパク質レベルにおいてみられる増加をもたらし得る。
〔実施例4:トランスクリプトームおよびセクレトームの研究のための材料および方法〕
(株)
すべてのNeurospora crassa株を、Fungal Genetics Stock Center(FGSC;ウェブページfgsc.net)から入手した(Tian et al., 2009における第1頁の、Supplemental Data, Dataset S1)。遺伝子欠失株を、N. crassaの機能ゲノムプロジェクト(N. crassa functional genomics project)に由来する(Dunlap et al., 2007)。Trichoderma reesei QM9414はMonika Schmoll博士(ウィーン工科大学)から頂いた。株を、2%の(w/v)の炭素源(Miscanthus,スクロースまたはAvicel(Sigma))を含んでいるVogel'sの塩(Vogel 1956)において成長させた。Miscanthus x giganteus(〜0.1mmに粉砕した茎)をイリノイ大学から頂いた。
(株)
すべてのNeurospora crassa株を、Fungal Genetics Stock Center(FGSC;ウェブページfgsc.net)から入手した(Tian et al., 2009における第1頁の、Supplemental Data, Dataset S1)。遺伝子欠失株を、N. crassaの機能ゲノムプロジェクト(N. crassa functional genomics project)に由来する(Dunlap et al., 2007)。Trichoderma reesei QM9414はMonika Schmoll博士(ウィーン工科大学)から頂いた。株を、2%の(w/v)の炭素源(Miscanthus,スクロースまたはAvicel(Sigma))を含んでいるVogel'sの塩(Vogel 1956)において成長させた。Miscanthus x giganteus(〜0.1mmに粉砕した茎)をイリノイ大学から頂いた。
(酵素活性測定)
細胞外タンパク質の総含有量を、Bio-Rad DC Protein Assayキット(Bio-Rad)を用いて測定した。培養上清におけるエンドグルカナーゼ活性を、azo-CMCキット(Megazyme SCMCL)を用いて測定した。β−グルコシダーゼ活性を、10倍に希釈した培養上清を、50mMの酢酸ナトリウム緩衝液(pH5.0)における500μMの4−ニトロフェニルβ−D−グルコピラノシドとともに40℃において10分にわたって混合することによって測定した。5%のw/vの炭酸ナトリウムを用いて反応を停止させ、400nmにおける吸光度を測定した。Avicerase活性を、2倍に希釈した培養上清を50mMの酢酸ナトリウム緩衝液(pH5.0)および5mg/mLのAvicelと40℃において混合することによって測定した。上清を、グルコースオキシダーゼ/ペルオキシダーゼによる共役酵素アッセイを用いてグルコース含有量について分析した。100mMの酢酸ナトリウム、pH5.0、10U/mLのホースラディッシュペルオキシダーゼ、10U/mLのグルコースオキシダーゼおよび1mMのo−ジアニシジンを含んでいる150μLのグルコース検出試薬に、50μLのAvicerase反応液を移した。540nmにおける吸光度を30分後に測定した。セロビオースの濃度を、Sporotrichum thermophile由来のセロビオースデヒドロゲナーゼ(CDH)による共役酵素アッセイを用いて測定した。これまでの研究報告書(Canevascini 1988)と同様にS. thermophileからCDHを単離した。125mMの酢酸ナトリウム(pH5.0)、250μMのジクロロフェノールインドフェノール、および0.03mg/mLのCDHを含んでいる250μLのセロビオース検出試薬に50μLのAvicerase反応液を移した。530nmにおける吸光度を10分後に測定した。
細胞外タンパク質の総含有量を、Bio-Rad DC Protein Assayキット(Bio-Rad)を用いて測定した。培養上清におけるエンドグルカナーゼ活性を、azo-CMCキット(Megazyme SCMCL)を用いて測定した。β−グルコシダーゼ活性を、10倍に希釈した培養上清を、50mMの酢酸ナトリウム緩衝液(pH5.0)における500μMの4−ニトロフェニルβ−D−グルコピラノシドとともに40℃において10分にわたって混合することによって測定した。5%のw/vの炭酸ナトリウムを用いて反応を停止させ、400nmにおける吸光度を測定した。Avicerase活性を、2倍に希釈した培養上清を50mMの酢酸ナトリウム緩衝液(pH5.0)および5mg/mLのAvicelと40℃において混合することによって測定した。上清を、グルコースオキシダーゼ/ペルオキシダーゼによる共役酵素アッセイを用いてグルコース含有量について分析した。100mMの酢酸ナトリウム、pH5.0、10U/mLのホースラディッシュペルオキシダーゼ、10U/mLのグルコースオキシダーゼおよび1mMのo−ジアニシジンを含んでいる150μLのグルコース検出試薬に、50μLのAvicerase反応液を移した。540nmにおける吸光度を30分後に測定した。セロビオースの濃度を、Sporotrichum thermophile由来のセロビオースデヒドロゲナーゼ(CDH)による共役酵素アッセイを用いて測定した。これまでの研究報告書(Canevascini 1988)と同様にS. thermophileからCDHを単離した。125mMの酢酸ナトリウム(pH5.0)、250μMのジクロロフェノールインドフェノール、および0.03mg/mLのCDHを含んでいる250μLのセロビオース検出試薬に50μLのAvicerase反応液を移した。530nmにおける吸光度を10分後に測定した。
(RNAの単離、マイクロアレイ分析、およびシグナルペプチド予測)
菌糸体を、ろ過によって回収し、液体窒素において瞬間冷凍した。トリゾールを用いて総RNAを単離した(Tian et al., 2007;Kasuga et al., 2005)。マイクロアレイハイブリダイゼーションおよびデータ分析は、これまでに説明されている通りである(Tian et al., 2007)。標準化した発現値をBAGEL(遺伝子発現レベルのベイズの解析、Bayesian analysis of gene expression levels)を用いて分析した(Townsend 2004;Townsend and Hartl 2002)。BAGELは、実験の各時点における各遺伝子について相対的な遺伝子発現レベルおよび信頼区間を推測するものである。シグナルペプチドを、シグナルP3プログラム(ウェブページcbs.dtu.dk/services/SignalP-3.0/)によって予想されている各タンパク質のN末端の70アミノ酸を用いて予想した。元のプロファイリングデータは、(ウェブページyale.edu/townsend/Links/ffdatabase/)において入手可能である。
菌糸体を、ろ過によって回収し、液体窒素において瞬間冷凍した。トリゾールを用いて総RNAを単離した(Tian et al., 2007;Kasuga et al., 2005)。マイクロアレイハイブリダイゼーションおよびデータ分析は、これまでに説明されている通りである(Tian et al., 2007)。標準化した発現値をBAGEL(遺伝子発現レベルのベイズの解析、Bayesian analysis of gene expression levels)を用いて分析した(Townsend 2004;Townsend and Hartl 2002)。BAGELは、実験の各時点における各遺伝子について相対的な遺伝子発現レベルおよび信頼区間を推測するものである。シグナルペプチドを、シグナルP3プログラム(ウェブページcbs.dtu.dk/services/SignalP-3.0/)によって予想されている各タンパク質のN末端の70アミノ酸を用いて予想した。元のプロファイリングデータは、(ウェブページyale.edu/townsend/Links/ffdatabase/)において入手可能である。
(タンパク質のゲル電気泳動)
特に断りがない限り、Criterionの4〜15%のTris−HClポリアクリルアミドゲルにロードする前に、濃縮していない培養上清を5×SDSローディング染色液および5分間にわたる煮沸によって処理した。クマシー染色液を染色のために用いた。
特に断りがない限り、Criterionの4〜15%のTris−HClポリアクリルアミドゲルにロードする前に、濃縮していない培養上清を5×SDSローディング染色液および5分間にわたる煮沸によって処理した。クマシー染色液を染色のために用いた。
(セクレトーム分析のためのトリプシンペプチドの調製)
10 kDa MWCO PESスピン濃縮器を用いて培養上清を濃縮した。タンパク質に結合しているセルロースを、リン酸膨潤セルロース(PASC)の添加によって培養上清から単離した。10mg/mLのPACSのうち5mLの上清を10mLの培養上清に加えた。4℃において5分にわたってインキュベートした後、混合物を遠心分離し、それから沈殿したPASCを20倍容積の100mM酢酸ナトリウム(pH5.0)を用いて洗浄した。PASCによる処理後の上清を、非結合画分として収集し、濃縮した。それから、36mgの尿素、5μLの1MのTris(pH8.5)、および5μLの100mMのDTTを、100μLの濃縮した培養上清またはタンパク質と結合させたPASCに加え、混合物を60℃において1時間にわたって加熱した。加熱後に、700μLの25mMの炭酸水素アンモニウムおよび140μLのメタノールを上記溶液に加え、それから、50mMの酢酸ナトリウム(pH5.0)における50μLの100μg/mLのトリプシンによって処理した。PASC結合タンパク質について、PASCを加熱後に遠心分離によって除去し、それから上清をトリプシンによって処理した。トリプシンを残したままで37℃において一晩にわたって反応させた。消化後、容積をスピードバック(speedvac)によって減らし、ミリQ水を用いて3回にわたって洗浄した。製造者の手順にしたがってOMIX微量抽出ピペットチップを用いることによって、サンプル中に残存している塩を除去した。
10 kDa MWCO PESスピン濃縮器を用いて培養上清を濃縮した。タンパク質に結合しているセルロースを、リン酸膨潤セルロース(PASC)の添加によって培養上清から単離した。10mg/mLのPACSのうち5mLの上清を10mLの培養上清に加えた。4℃において5分にわたってインキュベートした後、混合物を遠心分離し、それから沈殿したPASCを20倍容積の100mM酢酸ナトリウム(pH5.0)を用いて洗浄した。PASCによる処理後の上清を、非結合画分として収集し、濃縮した。それから、36mgの尿素、5μLの1MのTris(pH8.5)、および5μLの100mMのDTTを、100μLの濃縮した培養上清またはタンパク質と結合させたPASCに加え、混合物を60℃において1時間にわたって加熱した。加熱後に、700μLの25mMの炭酸水素アンモニウムおよび140μLのメタノールを上記溶液に加え、それから、50mMの酢酸ナトリウム(pH5.0)における50μLの100μg/mLのトリプシンによって処理した。PASC結合タンパク質について、PASCを加熱後に遠心分離によって除去し、それから上清をトリプシンによって処理した。トリプシンを残したままで37℃において一晩にわたって反応させた。消化後、容積をスピードバック(speedvac)によって減らし、ミリQ水を用いて3回にわたって洗浄した。製造者の手順にしたがってOMIX微量抽出ピペットチップを用いることによって、サンプル中に残存している塩を除去した。
(トリプシンペプチドの液体クロマトグラフィー)
超高性能液体クロマトグラフィー(UPLC)とタンデムに接続したタンデム質量分析装置を用いて、トリプシン消化したタンパク質を分析した。C18トラッピング(180μm×20mm)、分析(100μm×100mm)カラムおよび10μLのサンプルループを備えているnanoAcquity UPLC(Waters、Milford、MA)を用いてペプチドを分離した。溶液Aは0.1%の蟻酸/99.9%の水であり、溶液Bは0.1%の蟻酸/99.9%のアセトニトリル(v/v)であった。隔壁のキャップを用いて密閉した0.3mLのポリプロピレン製の分水栓バイアル(Wheaton Science, Millville, NJ)に入っているサンプル溶液を、分析前にnanoAcquity自動試料採取器にロードした。サンプルの注入(2μL、部分的なループ)後、トラッピングを3μL/分において100%のAによって5分にわたって行った。サンプル間の相互の汚染を避けるために、注入後、注射針を750μLの溶媒AおよびBのそれぞれを用いて洗浄した。溶出のプログラムは、500nL/分の流速において、55分間にわたる5%〜30%までのBの直線勾配、20分間にわたる40%までのBの直線勾配、0.33分間にわたる95%までのBの直線勾配、11.67分間にわたる95%のBの定組成状態、0.33分間にわたる1%までのBの直線勾配、および11.67分間にわたる1%のBの定組成状態から構成された。分析カラムおよびサンプル容器はそれぞれ35℃および8℃に維持した。
超高性能液体クロマトグラフィー(UPLC)とタンデムに接続したタンデム質量分析装置を用いて、トリプシン消化したタンパク質を分析した。C18トラッピング(180μm×20mm)、分析(100μm×100mm)カラムおよび10μLのサンプルループを備えているnanoAcquity UPLC(Waters、Milford、MA)を用いてペプチドを分離した。溶液Aは0.1%の蟻酸/99.9%の水であり、溶液Bは0.1%の蟻酸/99.9%のアセトニトリル(v/v)であった。隔壁のキャップを用いて密閉した0.3mLのポリプロピレン製の分水栓バイアル(Wheaton Science, Millville, NJ)に入っているサンプル溶液を、分析前にnanoAcquity自動試料採取器にロードした。サンプルの注入(2μL、部分的なループ)後、トラッピングを3μL/分において100%のAによって5分にわたって行った。サンプル間の相互の汚染を避けるために、注入後、注射針を750μLの溶媒AおよびBのそれぞれを用いて洗浄した。溶出のプログラムは、500nL/分の流速において、55分間にわたる5%〜30%までのBの直線勾配、20分間にわたる40%までのBの直線勾配、0.33分間にわたる95%までのBの直線勾配、11.67分間にわたる95%のBの定組成状態、0.33分間にわたる1%までのBの直線勾配、および11.67分間にわたる1%のBの定組成状態から構成された。分析カラムおよびサンプル容器はそれぞれ35℃および8℃に維持した。
(質量分析)
四重極子飛行時間型質量分析計(Q-Tof Premier、Waters)のナノフローイオン源に搭載させたNanoEaseナノエレクトロスプレーイオン化(ナノESI)放射源にカラムを接続した。ナノESI源のパラメーターは以下の通りである:2.3kVのナノESIキャピラリー電圧、0.15mbarの噴霧気体(窒素)圧、30Vのサンプルコーン電圧、5Vの抽出コーン電圧、3Vのイオン導入圧力、80℃の電源ブロック温度。分析装置のTは“V”モードにおいて操作した。これらの条件において1.0×104の質量分解能(m/z=771において測定した)を通常に達成したが、これは、本研究において1価または多価の電荷を有しているペプチドイオンの同位体分布の分析にとって十分である。このように、イオンの質量および電荷を独立して決定し得た。すなわち、イオン電荷をm/zスペクトルにおける隣接している同位体ピークの間隔の逆数から決定した。分析の直前に蟻酸ナトリウムを用いて外的に質量補正を行った。0.95秒間のスキャン積分および0.05秒間のスキャンごとの間隔を用いて、m/z=450〜1800の範囲にわたる陽イオンモードにおいて測量スキャンを得た。タンデム質量分析(MS/MS)に関して、データ従属モードにおいて、35カウント/秒間(cps)の強度閾値を超えている5までの前駆イオンを、各測量スキャンから選択した。MS/MSに関して、2+、3+、4+、5+、および6+の電荷状態の前駆イオンを選択するために、実時間の脱同位体化(real-time deisotoping)および電荷状態認識を用いた。衝突活性化分離(CAD)のための衝突エネルギーを、質量および所定の前駆イオンの電荷状態に基づいて自動的に選択した。0.95秒間のスキャン積分および0.05秒間のスキャンごとの間隔を用いて、m/z=50〜2500の範囲にわたってMS/MSスペクトルを得た。累積MS/MSスペクトルにおいて30000cpsの最小総イオン電流(TIC)を達成するために、最大3秒にわたってイオンを分裂させた。不要なMS/MS測定の発生の回避を目的として、前に分析した前駆イオンの再選択を排除するために、180秒間、0.25m/zユニットの排除幅にわたって、リアルタイム排除を用いた。
四重極子飛行時間型質量分析計(Q-Tof Premier、Waters)のナノフローイオン源に搭載させたNanoEaseナノエレクトロスプレーイオン化(ナノESI)放射源にカラムを接続した。ナノESI源のパラメーターは以下の通りである:2.3kVのナノESIキャピラリー電圧、0.15mbarの噴霧気体(窒素)圧、30Vのサンプルコーン電圧、5Vの抽出コーン電圧、3Vのイオン導入圧力、80℃の電源ブロック温度。分析装置のTは“V”モードにおいて操作した。これらの条件において1.0×104の質量分解能(m/z=771において測定した)を通常に達成したが、これは、本研究において1価または多価の電荷を有しているペプチドイオンの同位体分布の分析にとって十分である。このように、イオンの質量および電荷を独立して決定し得た。すなわち、イオン電荷をm/zスペクトルにおける隣接している同位体ピークの間隔の逆数から決定した。分析の直前に蟻酸ナトリウムを用いて外的に質量補正を行った。0.95秒間のスキャン積分および0.05秒間のスキャンごとの間隔を用いて、m/z=450〜1800の範囲にわたる陽イオンモードにおいて測量スキャンを得た。タンデム質量分析(MS/MS)に関して、データ従属モードにおいて、35カウント/秒間(cps)の強度閾値を超えている5までの前駆イオンを、各測量スキャンから選択した。MS/MSに関して、2+、3+、4+、5+、および6+の電荷状態の前駆イオンを選択するために、実時間の脱同位体化(real-time deisotoping)および電荷状態認識を用いた。衝突活性化分離(CAD)のための衝突エネルギーを、質量および所定の前駆イオンの電荷状態に基づいて自動的に選択した。0.95秒間のスキャン積分および0.05秒間のスキャンごとの間隔を用いて、m/z=50〜2500の範囲にわたってMS/MSスペクトルを得た。累積MS/MSスペクトルにおいて30000cpsの最小総イオン電流(TIC)を達成するために、最大3秒にわたってイオンを分裂させた。不要なMS/MS測定の発生の回避を目的として、前に分析した前駆イオンの再選択を排除するために、180秒間、0.25m/zユニットの排除幅にわたって、リアルタイム排除を用いた。
(質量分析のデータ分析)
トリプシン消化されたタンパク質のLC−MS/MS分析から得られたデータを、ProteinLynx Global Serverソフトウェア(version 2.3、Waters)を用いて処理した。このソフトウェアは、質量スペクトルおよびMS/MSスペクトルの、バックグランウド減算(閾値35%および5次多項式)、平滑化(10回のSavitzky-Golay2、3チャンネル以上)、および重心化(上位80%の各ピークおよび半分の高さの4チャンネルにおける最小ピーク幅)を行うものであった。処理したデータをN. crassaのデータベース(Broad Institute)に対して、次の基準を用いて調べた:5つまでの開裂不良を有しているトリプシン断片、前駆イオン質量の50ppmの誤差、断片イオン質量の0.1Daの誤差、および種々の翻訳後修飾(N末端およびLys側鎖のカルバミル化、Metの酸化、ならびにSer/Thrの脱水)。ペプチドをMS/MSスペクトルに割り当てるために、同じ組に由来する少なくとも3つの連続した断片イオン(すなわちb型またはy型の断片イオン)の同定を必要とした。一義的にペプチドを同定する断片イオンの存在を確認するために、MS/MSスペクトルを手動で検定した。
トリプシン消化されたタンパク質のLC−MS/MS分析から得られたデータを、ProteinLynx Global Serverソフトウェア(version 2.3、Waters)を用いて処理した。このソフトウェアは、質量スペクトルおよびMS/MSスペクトルの、バックグランウド減算(閾値35%および5次多項式)、平滑化(10回のSavitzky-Golay2、3チャンネル以上)、および重心化(上位80%の各ピークおよび半分の高さの4チャンネルにおける最小ピーク幅)を行うものであった。処理したデータをN. crassaのデータベース(Broad Institute)に対して、次の基準を用いて調べた:5つまでの開裂不良を有しているトリプシン断片、前駆イオン質量の50ppmの誤差、断片イオン質量の0.1Daの誤差、および種々の翻訳後修飾(N末端およびLys側鎖のカルバミル化、Metの酸化、ならびにSer/Thrの脱水)。ペプチドをMS/MSスペクトルに割り当てるために、同じ組に由来する少なくとも3つの連続した断片イオン(すなわちb型またはy型の断片イオン)の同定を必要とした。一義的にペプチドを同定する断片イオンの存在を確認するために、MS/MSスペクトルを手動で検定した。
(定量的RT−PCR)
Qiagenからの試薬(SYBR-green RT-PCR kit(Cat No. 204243))を用いて、ABI7300においてRT−PCRを行った。CBHI(NCU07340)用プライマー:順方向5’-ATCTGGGAAGCGAACAAAG-3’(配列番号16)および逆方向5’-TAGCGGTCGTCGGAATAG-3’(配列番号17)。CBHII(NCU09680)用プライマー;順方向5’-CCCATCACCACTACTACC-3’(配列番号18)および逆方向5’-CCAGCCCTGAACACCAAG-3’(配列番号19)。アクチンを標準化のための対照として用いた。アクチン用プライマー;順方向5’-TGA TCT TAC CGA CTA CCT -3’(配列番号20)および逆方向5’-CAG AGC TTC TCC TTG ATG -3’(配列番号21)。Dementhon et al., (2006)にしたがって定量的RT−PCRを行った。
Qiagenからの試薬(SYBR-green RT-PCR kit(Cat No. 204243))を用いて、ABI7300においてRT−PCRを行った。CBHI(NCU07340)用プライマー:順方向5’-ATCTGGGAAGCGAACAAAG-3’(配列番号16)および逆方向5’-TAGCGGTCGTCGGAATAG-3’(配列番号17)。CBHII(NCU09680)用プライマー;順方向5’-CCCATCACCACTACTACC-3’(配列番号18)および逆方向5’-CCAGCCCTGAACACCAAG-3’(配列番号19)。アクチンを標準化のための対照として用いた。アクチン用プライマー;順方向5’-TGA TCT TAC CGA CTA CCT -3’(配列番号20)および逆方向5’-CAG AGC TTC TCC TTG ATG -3’(配列番号21)。Dementhon et al., (2006)にしたがって定量的RT−PCRを行った。
〔実施例5:トランスクリプトームおよびセクレトームの研究の考察〕
植物バイオマスの分解は、多くの異なった酵素活性の産生を必要とするが、この酵素活性は、利用可能な植物材料の種類および複合度によって調節される(図9)(Bouws et al., 2008)。セルロース分解性の真菌による植物の細胞壁の分解における最初の体系的な分析についてここに説明する。当該分析は、トランスクリプトーム分析、セクレトーム分析、および変異体分析を包含している。プロファイリングデータは、N. crassaをMiscanthusまたはAvicelに基づく成長によって試験したとき、多数の細胞外および細胞内タンパク質を協調的に発現することを示した(図9)。セルロース性基質に基づく成長の間に最も多く発現された遺伝子の多くは、植物の細胞壁の多糖の代謝に関与すると予想タンパク質(その多くがMS分析によって同定された)をコードしている。糸状菌のゲノム比較は、種々の数(N. crassaと近縁の肥料分解種であるT. reeseiにおける10(Martinez et al., 2008)からPodospora anserinaにおける60(Espagne et al., 2008)まで)の予想されているセルラーゼを含む多数のグリコシルヒドロラーゼ(〜200)を示す。これらの結果および白腐担子菌のPhanerochaete chrysosporiumにおける近年のトラスクリプトーム/セクレトーム研究(Wymelenberg et al., 2009)の間の比較は、両方の種が純粋なセルロースに基づいて成長したときに、調節遺伝子(18の遺伝子)および分泌タンパク質(2のタンパク質)におけるわずかな重複を示した。これらのデータは、異なる真菌が植物の細胞壁の分解のために異なる遺伝子セットを利用することを示唆する。しかし、両研究に共通していた1つの局面は、性質決定されていない、セルロース分解に関与する非常に多くの遺伝子/タンパク質であった。セルロース分解性の他の真菌(P. chrysosporiumが挙げられる)は、N. crassaにおいて直ちに利用可能な遺伝子ツールおよび分子ツールを有していない。N. crassaにおいて利用可能な機能的なゲノムツールを用いて、植物の細胞壁の分解酵素系の機能および重複の両方を、リグノセルロースバイオマス由来の液体燃料の工業製品に最適な酵素混合物を作製するために検討し得る。
植物バイオマスの分解は、多くの異なった酵素活性の産生を必要とするが、この酵素活性は、利用可能な植物材料の種類および複合度によって調節される(図9)(Bouws et al., 2008)。セルロース分解性の真菌による植物の細胞壁の分解における最初の体系的な分析についてここに説明する。当該分析は、トランスクリプトーム分析、セクレトーム分析、および変異体分析を包含している。プロファイリングデータは、N. crassaをMiscanthusまたはAvicelに基づく成長によって試験したとき、多数の細胞外および細胞内タンパク質を協調的に発現することを示した(図9)。セルロース性基質に基づく成長の間に最も多く発現された遺伝子の多くは、植物の細胞壁の多糖の代謝に関与すると予想タンパク質(その多くがMS分析によって同定された)をコードしている。糸状菌のゲノム比較は、種々の数(N. crassaと近縁の肥料分解種であるT. reeseiにおける10(Martinez et al., 2008)からPodospora anserinaにおける60(Espagne et al., 2008)まで)の予想されているセルラーゼを含む多数のグリコシルヒドロラーゼ(〜200)を示す。これらの結果および白腐担子菌のPhanerochaete chrysosporiumにおける近年のトラスクリプトーム/セクレトーム研究(Wymelenberg et al., 2009)の間の比較は、両方の種が純粋なセルロースに基づいて成長したときに、調節遺伝子(18の遺伝子)および分泌タンパク質(2のタンパク質)におけるわずかな重複を示した。これらのデータは、異なる真菌が植物の細胞壁の分解のために異なる遺伝子セットを利用することを示唆する。しかし、両研究に共通していた1つの局面は、性質決定されていない、セルロース分解に関与する非常に多くの遺伝子/タンパク質であった。セルロース分解性の他の真菌(P. chrysosporiumが挙げられる)は、N. crassaにおいて直ちに利用可能な遺伝子ツールおよび分子ツールを有していない。N. crassaにおいて利用可能な機能的なゲノムツールを用いて、植物の細胞壁の分解酵素系の機能および重複の両方を、リグノセルロースバイオマス由来の液体燃料の工業製品に最適な酵素混合物を作製するために検討し得る。
本研究において、N. crassaにおけるセロビオヒドロラーゼ(I)(CBHI)が、AvicelまたはMiscanthusに基づく成長期間に最も多く産生される細胞外タンパク質であること、およびこの遺伝子の欠失がセルロース性基質において最も深刻な成長不全を引き起こすことを見出した。また、これらの結果はT. reeseiにおいて報告されたもの(Suominen et al., 1993, Seiboth et al., 1997)と類似する。セロビオヒドロラーゼ(II)の欠失は、セルロース性基質に基づく成長不良を引き起こすが、CBH(I)よりかなり程度が小さく、N. crassaにおけるエキソグルカナーゼ活性がCBH(I)にほとんどが由来すること、および他のCBHがCBH(I)の欠失を補わないことを示唆している。ここで、セルロース性基質に基づく成長期間に最も多く産生される3つのエンドグルカナーゼが、NCU05057、NCU00762、およびNCU07190によってコードされているタンパク質であることが示されていた。これらのタンパク質は、それぞれ、エンドグルカナーゼEG1、EG2、およびEG6と相同性を有している。これらの遺伝子の欠失は、Avicelに基づく成長に影響を与えなかったが、分泌タンパク質のレベルおよびエンドグルカナーゼ活性に差異が認められた。意外にも、ΔNCU05057株において、細胞外タンパク質(特にCBH(I))のレベルは非常に高く、結晶性セルロースにおける野生型の成長特性を維持するために、この変異体は、他のセルラーゼの産生を促進しているか、またはNCU05057の触媒作用の生成物がセルラーゼ産生を抑制し得ることを示唆する。N. crassaにおけるエンドグルカナーゼはいずれも、純粋なセルロースに基づく成長に必要ではなく、異なるエンドグルカナーゼは、重複した基質特異性を有していると結論付けた。
グリコシドヒドロラーゼファミリー61酵素は、T. reeseiと比較してN. crassaにおいて非常に拡大されている(Martinez et al., 2008)。これらの酵素は生物学的機能が十分には明確となっていないが、セルロースを分解できる菌におけるそれらの普遍的な保存性および存在量は植物の細胞壁の代謝における重要な役割を示唆している。ここで、14のGH61のうちの10個に関する遺伝子はN. crassaのトランスクリプトームにおいて同定され、セルロースのバイオマスに基づく成長期間にこれらの酵素が利用されることを示唆した。試験した4のGH61欠失株は、分泌タンパク質のレベル、エンドグルカナーゼ、および全セルラーゼ活性において、野生型と比較してほんのわずかな違いを示すだけであった。しかし、追加のGH61変異体の分析およびN. crassaにおける交雑による複数の変異を有している株の作製可能性は、重複性に取り組み、このファミリーの機能分析を促進し得る。
予想されているセルラーゼ遺伝子に加え、ヘミセルラーゼをコードしている遺伝子、炭水化物エステラーゼ、β−グルコシダーゼ、β−キシロシダーゼ、および炭水化物に対する活性を有していると予想されている他のタンパク質は、MiscanthusおよびAvicelの両方からのN. crassaのトランスクリプトームにおいて同定された。Avicelがヘミセルロース成分を含んでいないという事実は、セルロースがおそらくN. crassaにおいて植物の細胞壁の分解酵素をコードしている遺伝子の一次誘導物質であることを示唆する。しかし、いくつかのヘミセルラーゼおよび炭水化物エステラーゼをコードしている遺伝子は、Miscanthusに基づく成長期間にのみ発現する。同様に、T. reeseiおよびAspergillus nigerなどのセルロースを分解できる他の菌において、いくつかのセルラーゼおよびヘミセルラーゼをコードしている遺伝子は協同的に調節され、一方、他は別々に調節される(Stricker et al., 2008)。予想した通り、セルラーゼでない遺伝子の欠失は、NCU05137およびgh3−4を除いて、Avicelに基づく成長またはセルラーゼ活性にほとんど影響を与えなかった。より多くのタンパク質が分泌されたΔNCU05137株は、野生型よりも、高いセルラーゼ活性を有しており、高いcbh−1(CBH(I))およびgh6−2(CBH(II))の発現を示した。NCU05137は、機能既知のタンパク質と相同性を有していない分泌される仮定のタンパク質をコードしているが、セルラーゼを分解できる他の菌において高度に保存されている(図2;E値0.0)。また、NCU05137は、多くの真正細菌において、より遠縁の相同物(同様に機能未知)を有している。NCU05137のタンパク質産物は、N. crassaのセルラーゼ遺伝子発現の誘導と関連するシグナル伝達の過程を妨害し得る(図9)。同様に、gh3−4(NCU04952)における変異体もCBH(I)活性が増加した。この遺伝子に欠失はPNPGアーゼ活性を完全に取り除き、Δgh3−4培養ろ過物を用いたインビトロにおけるセルラーゼアッセイにおいてセロビオースを蓄積した。すべてのデータが、N. crassaにおいて、NCU04952が一次的な細胞外β−グルコシダーゼをコードしていることを総合的に示唆した。
セルロースおよびヘミセルロースの細胞外分解は、細胞内へ次いで輸送される可溶性の炭水化物の形成を引き起こす(図9)。本研究において、パーミアーゼ/トランスポータをコードしている10の遺伝子を、N. crassaがMiscanthusまたはAvicelに基づいて成長した際に顕著に増加した発現を示すことを同定しており、それは植物の細胞壁の分解産物の細胞への輸送に関係していることを示唆している。インビトロにおけるセルラーゼおよびヘミセルラーゼによる主な分解産物は、セロビオース、グルコース、キシロビオース、およびキシロースである。これらのトランスポータのいくつかは、機能的に重複し得るか、またはオリゴ糖を輸送し得る。これらの推定トランスポータの機能をさらに調べた(実施例7〜9を参照)。N. crassaトランスポータの非相同的発現によってオリゴ糖を輸送できる下流の構成を処理している株は、バイオマスの加水分解産物の工業的発酵を改良し得る。これらのトランスポータまたは輸送し得るものはいずれも、任意の糸状菌において、分子または機能レベルにおいて性質決定がなされていない。
MiscanthusおよびAvicelに基づく成長期間に発現レベルの増加が示された多くの遺伝子は、セルロースを分解できる他の菌において保存されている機能未知のタンパク質をコードしている。わずか16株の表現型を評価することによって、セルラーゼ活性に顕著に影響を与える機能未知のタンパク質をコードしている遺伝子における変異体が同定された。公知の遺伝学およびN. crassaにおける機能的ゲノム資源の利用可能性は、N. crassaを、これらのタンパク質の生物学的機能、同様にセルラーゼおよびセミセルラーゼ産物の調節形態の決定するため、ならびに植物の細胞壁の分解に関係する細胞外酵素間の重複および相乗作用を分析するための理想的なモデル生物にする。
〔実施例6:Miscanthusに基づく成長期間に上方制御された遺伝子の変異体のスクリーニング〕
転写のプロファイリング実験において同定された別の遺伝子を分析するために、Miscanthusに基づいて16時間にわたって成長させたNeurosporaにおいて上方制御された188の遺伝子の変異体の表現型を分析した(実施例1を参照)。各遺伝子のノックアウト変異体をVogel's最少培地に基づいて10〜14日にわたって成長させた。2mLのddH2Oを用いて分生子を回収し、250mLフラスコに入っている100mL培地に、1mLあたり106分生子の濃度において播種した。3つの異なる炭素源のうち1つを各フラスコに加えた:2%のスクロース、2%のAvicel、または2%のMiscanthus(カリフォルニア州バークリーにあるカリフォルニア大学のカルビン研究室からの1mmの粒子)。培養物は220rpmの振とうをしながら、25℃において4日にわたって成長させた。
転写のプロファイリング実験において同定された別の遺伝子を分析するために、Miscanthusに基づいて16時間にわたって成長させたNeurosporaにおいて上方制御された188の遺伝子の変異体の表現型を分析した(実施例1を参照)。各遺伝子のノックアウト変異体をVogel's最少培地に基づいて10〜14日にわたって成長させた。2mLのddH2Oを用いて分生子を回収し、250mLフラスコに入っている100mL培地に、1mLあたり106分生子の濃度において播種した。3つの異なる炭素源のうち1つを各フラスコに加えた:2%のスクロース、2%のAvicel、または2%のMiscanthus(カリフォルニア州バークリーにあるカリフォルニア大学のカルビン研究室からの1mmの粒子)。培養物は220rpmの振とうをしながら、25℃において4日にわたって成長させた。
表10は、セルラーゼ活性およびMiscanthusまたはAvicelに基づく成長において野生型と比較して顕著な違いを示した変異体の表現型を列挙している。MiscanthusまたはAvicelに基づく成長を、“+”得点法を用いて肉眼によって評価した。培養上清における総タンパク質を、Bradfordアッセイ(100μlの上清を900μlBradford染色液へ入れる)によって測定した。エンドグルカナーゼ活性をMegazyme製のAzo-CMCキットを用いて測定し、野性型と比較した変異体におけるエンドグルカナーゼ活性のパーセンテージとして表10に示した。全セルラーゼ活性を、実施例4に記載したように上清におけるセロビオースのレベルを検出することによって測定した。結果を野生型のパーセンテージとして表10に示した。
〔実施例7:トランスポータ遺伝子のさらなる分析〕
実施例1に記載したように、予想されている糖輸送タンパク質をコードしている10の遺伝子は、Neurospora をMiscanthusおよびAvicelに基づいて成長された際に発現レベルの増加を示した:NCU00801、NCU00988、NCU01231、NCU04963、NCU05519、NCU05853、NCU05897、NCU06138、NCU08114およびNCU10021。これらの遺伝子のうち9個についての欠失株が、Fungal Genetics Stock Centerから入手できた。NCU10021の欠失株は入手できなかった。
実施例1に記載したように、予想されている糖輸送タンパク質をコードしている10の遺伝子は、Neurospora をMiscanthusおよびAvicelに基づいて成長された際に発現レベルの増加を示した:NCU00801、NCU00988、NCU01231、NCU04963、NCU05519、NCU05853、NCU05897、NCU06138、NCU08114およびNCU10021。これらの遺伝子のうち9個についての欠失株が、Fungal Genetics Stock Centerから入手できた。NCU10021の欠失株は入手できなかった。
NCU05853、NCU05897、またはNCU08114の欠失株は、MiscanthusまたはAvicelに基づく成長不全を示す、および/もしくは、セルラーゼ酵素が欠失している株となった(実施例6;表10を参照)。ΔNCU05853は、野生型と比べて、Avicelに基づく成長の減少およびエンドグルカナーゼ活性の低下を示した。ΔNCU05897は、野生型と比べて、Avicelに基づく成長の減少およびエンドグルカナーゼ活性の低下を示した。また、ΔNCU08114は、野生型と比べて、Avicelに基づく成長の減少およびセロビオースのレベルの低下を示した。特に、Sporotrichum thermophile(別の糸状菌)の発現分析との比較において、S. thermophileをAvicelに基づいて成長させた際に、グルコースと比べて、NCU05853(ST8454)およびNCU08114(ST5194)の相同物も上方制御され(実施例8、表11を参照)、セロビオース利用におけるそれらの重要性をさらに示した。
各予想されているトランスポータの基質の同一性を絞り込むために、NCU05853またはNCU08114の欠失変異を有している株をグルコース、キシロース、セロビオース、キシランおよびAvicelに基づいて培養した(表12)。培養培地は2%の炭素源を足したVogel's最少培地から構成された。両方の変異体は、Avicelに基づく成長の大きな減少を示したが、キシラン、グルコース、キシロース、またはセロビオースにおいては示さなかった。
ヘミセルロースの利用におけるこれらのトランスポータの役割を調べるために、Neurosporaをキシランに基づいて成長させた際に、10のトランスポータ遺伝子の発現を試験した。2%(w/v)のキシランを含んでいるVogel'sの塩に基づいて成長させたことを除いて、株を実施例4に記載した方法を用いた。10個すべてのトランスポータの発現はキシランに基づく成長期間に上方制御され(表13)、それらがセルロース分解に由来する糖(例えば、セロビオース、グルコース、セロ−オリゴ糖)だけでなく、ヘミセルロース分解に由来する糖(例えば、キシロビオース、キシロース、アラビノース、キシロオリゴ糖)を輸送し得ることを示唆した。変異体の成長結果および発現分析は、少なくとも2つの予想されているトランスポータ(NCU05853およびNCU08114)が二糖(セロビオース、キシロビオース)および/またはオリゴ糖(セロデキストリン)を輸送し得ることを示唆した。
〔実施例8:種々の炭素源に基づく成長期間におけるN. crassaトランスポータのSporotrichum thermophile相同物の発現分析〕
異なる糸状菌由来の相同遺伝子の発現を比較するために、Sporotrichum thermophileの発現プロファイルを、グルコース、Avicel、または綿のに基づいて成長させた培養物から分析した。グルコース、Avicel、または綿を含んでいる最少培地に基づいて16〜30時間にわたって成長させた培養物からcDNAを単離した。
異なる糸状菌由来の相同遺伝子の発現を比較するために、Sporotrichum thermophileの発現プロファイルを、グルコース、Avicel、または綿のに基づいて成長させた培養物から分析した。グルコース、Avicel、または綿を含んでいる最少培地に基づいて16〜30時間にわたって成長させた培養物からcDNAを単離した。
まず、S. thermophileゲノムにおける、Neurosporaトランスポータタンパク質の相同物を同定するために、BLASTを用いて、各Neurosporaの配列をS. thermophileタンパク質のデータベースと比較した。次いで、この方法によってみつけたS. thermophileタンパク質の配列を、BLASTを用いて、Neurosporaタンパク質のデータベースを比較した。これらの結果を図10に列挙している。同定された推定NeurosporaトランスポータのすべてのS. thermophile相同物についてのアミノ酸配列を配列番号22〜32にみつけられる。
次いで、S. thermophile相同物の発現プロファイルを試験した。データを表11に示す。1列目は、Joint Genome Institute S. thermophile集団からのS. thermophile遺伝子名に相当する。2列目は、Neurosporaにおける最も近縁の推定トランスポータについてのNCU番号に相当する。3列目は、ヌクレオチドにおけるS. thermophile遺伝子の遺伝子長に相当する。4から6列目は、炭素源として2%のグルコース、Avicel、また脱脂綿を補ったVogel's最少培地に基づく成長期間の発現レベル(絶対的な発現レベルと比較可能な読み取りの数)に相当する。7から9列目は、標準化した発現データ(データセットにおける全読み取りによって分割された読み取りの♯)に相当する。最後の2列は、Avicel/グルコースまたは綿/グルコースの比とした、各遺伝子についての相対的な発現レベルのデータに相当する。NCU05853、NCU08114、およびNCU00801の相同物は、Avicelおよび綿の両方に基づいて成長させた場合に上方制御された。NCU06138の相同物は、綿に基づいて成長させた場合に上方制御され、NCU04963の相同物は、Avicelに基づいて成長させた場合に上方制御された。これらのデータは、推定トランスポータNCU05853、NCU08114、NCU00801、NCU06138、およびNCU04963がセルロースの利用に重要であるという、さらなる支持を提供する。
〔実施例9:セロデキストリントランスポータの同定および分析〕
純粋なセルロースに基づいて成長させた場合、N. crassaは、細胞内β−グルコシダーゼだけでなく、7の主要輸送促進体スーパーファミリー糖トランスポータの転写が増加することが示された(実施例1;Tian et al., PNAS, 2009のSupplemental Data, Dataset S1の第6頁を参照)。特に、このセットからの各トランスポータを欠いているノックアウト株は結晶性セルロースに基づいてより遅く成長し、セルロースを加水分解できる条件下において、それらがセロオリゴ糖の吸収において直接役割を果たし得ることを示唆する(実施例7;表10、12)。例えば、NCU08114の欠失はN. crassaの成長の深刻な遅滞(図11)、およびN. crassaのセロビオース消費の減少(図12〜13)を引き起こした。本実施例において、トランスポータ遺伝子NCU00801/cbt1およびNCU08114/cbt2を、セロデキストリンのトランスポータをコードすることをさらに分析および同定した。
純粋なセルロースに基づいて成長させた場合、N. crassaは、細胞内β−グルコシダーゼだけでなく、7の主要輸送促進体スーパーファミリー糖トランスポータの転写が増加することが示された(実施例1;Tian et al., PNAS, 2009のSupplemental Data, Dataset S1の第6頁を参照)。特に、このセットからの各トランスポータを欠いているノックアウト株は結晶性セルロースに基づいてより遅く成長し、セルロースを加水分解できる条件下において、それらがセロオリゴ糖の吸収において直接役割を果たし得ることを示唆する(実施例7;表10、12)。例えば、NCU08114の欠失はN. crassaの成長の深刻な遅滞(図11)、およびN. crassaのセロビオース消費の減少(図12〜13)を引き起こした。本実施例において、トランスポータ遺伝子NCU00801/cbt1およびNCU08114/cbt2を、セロデキストリンのトランスポータをコードすることをさらに分析および同定した。
各トランスポータの機能をそれぞれ分析するために、セロビオースがS. cerevisiaeによって触媒されず、その細胞質に蓄積されないという事実を利用した(図14)。セロビオースが単独の炭素源として与えられる場合に、細胞内β−グルコシダーゼと共役した機能性セロビオーストランスポータの発現がS. cerevisiaeを成長させ得ることが考えられた。緑色蛍光タンパク質(GFP)と融合したトランスポータNCU00801またはNCU08114、および推定細胞内β−グルコシダーゼNCU00130を発現するよう酵母株を処理した。両方のトランスポータが発現し、細胞質膜に正確に配置された(図15)。NCU00801またはNCU08114を発現している株は、それぞれ0.0341hr−1および0.0131hr−1の比成長速度において酵母を成長させた(図16A)。これらの成長速度は、それぞれグルコースに基づく成長速度の30%および12%に当たる(図17)。推定細胞内β−グルコシダーゼのみを発現している株が0.0026hr−1の速度において成長し(図16A)、大規模培養においては成長しなかった(図18)ため、セロビオースからグルコースへの細胞内加水分解、続く輸送によって成長を説明し得ない。これらの観察に基づき、NCU00801およびNCU08114(CBT1およびCBT2を名付けられた)は、セロビオーストランスポータとして機能すると結論付けた。
トランスポータ機能を直接分析するために、酵母細胞への[3H]−セロビオースの吸収を測定した。CBT1およびCBT2の両方が、それぞれ4.0±0.3μMおよび3.2±0.2μMのKm値を有している高い親和性セロビオーストランスポータであることがわかった(図19)。CBT1の発現標準化したVmaxは、CBT2のものの2.2倍であり、酵母の成長アッセイにおいてみられた違いを説明する事実であった。特に、セロビオースより長いセロデキストリン分子がcbt1およびcbt2を発現している酵母の成長を支えた(図20;図16B)。これは、セロデキストリン分子がCBT1およびCBT2によって輸送されることを示唆している。同様にして、CBT1およびCBT2によるセロビオース輸送は過剰のセロトリオースによって阻害され、CBT1活性もセロトリオースによって阻害された(図21)。その上、精製において、β−グルコシダーゼ、NCU00130(図22)は、セロビオース、セロトリオース、およびセロテトラオースを加水分解することがわかった(図16C)。
相同分子種cbt1およびcbt2は、真菌界に広く行き渡っていることを同定し、見出した(図23)。最近の発現データによって、真菌および植物の間における種々の相互作用に対するそれらの重要性が示されている。例えば、子嚢菌のTuber melanosporumまたは担子菌のLaccaria bicolorが根の先端と共生的に相互作用して、外性菌根を形成するとき、cbt1の相同分子種は両方において上方制御される(Martin et al., 2010)。同様に、腐生菌のAspergillus oryzae(Noguchi et al., 2009)、Postia placenta(Vanden Wymelenberg et al., 2010)およびPhanerochaete chrysosporium(Vanden Wymelenberg et al., 2010)は、植物の壁の材料と接触すると、cbt2の相同分子種を上方制御する。一部の酵母(例えば、Kluveromyces lactisおよびPichia stipitis)は、セロビオースに基づいて成長し(Freer, 1991; Preez et al., 1986)、セロビオース輸送がClavispora lusitaniaeにおいて報告されている(Freer and Greene 1990)。これらの酵母のすべては、cbt1、cbt2または両方の相同分子種を含んでいることが、本研究において決定された(方法については後の記載を参照)。セロビオース輸送はHypocrea jecorina(Trichoderma reesei)において観察されている。しかし、トランスポータは同定されていないので、この活性がcbt1またはcbt2の相同分子種に帰せられ得るか否かは明らかではない(Kubicek et al., 1993)。
セルロース分解性の真菌によるセロビオース輸送の利用は、それらがセルロースに基づく最適な成長に必須であることを示唆している。セルロースの異化作用が酵母のエタノール産生を向上させ得るか否かを試験するために、以上において構築された酵母株を発酵条件下において成長させた。わずかな至適化をともなって、N. crassaから移した完全なセロビオース異化経路を有している酵母は、86%の理論値(Bai et al., 2008)である0.47のエタノールの収率を有して、セロビオースをエタノールへと効率的に発酵させると示された(図24A)。これは、90〜93%のグルコースから得られた工業的な収率に匹敵した(Basso et al., 2008)。セロビオースに対するCBT1およびCBT2の高い親和性は、S. cerevisiaeのヘキソーストランスポータに匹敵し(Reifenberger et al., 1997)、細胞外のβ−グルコシダーゼと報告された(Chauve et al., 2010)。例えば、セロビオース/セロデキストリンの輸送は、グルコースへのセロビオースの完全な加水分解に対する要求性を低下させ、セルロース分解性の酵素のセロビオース媒介性の抑制を低減させ、グルコース依存性の生物による混入の危険性を下げる。実際に、セロビオース/セロデキストリンの輸送系を発現している酵母は、セロビオースおよびグルコースの両方の定常状態における濃度を低下させることおよびエタノールの産生速度を上昇させることによって、SSF反応の効率を顕著に向上させた(図24B、C)。
セルロースからの生物燃料の製造は、改良された宿主細胞による燃料の生成と協調した、糖への植物バイオマスの効率的かつ経済的な脱重合を必要とする(Kumar et al., 2008)。セルロース分解性の真菌は、植物バイオマスに基づく最適な成長のためにセロ−オリゴ糖輸送経路を利用することが示された。さらに、酵母におけるこれらの経路の再構築は、トランスポータおよび細胞内のセロ−オリゴ糖ヒドロラーゼによって構成されている最小の経路を用いて、セロビオースの異化を向上させるために、基本単位の様式においてそれらが移されることを明らかにした(図25)。酵母および他の生物の生物燃料を生成する株におけるセロデキストリン輸送の利用は、セルロース性の生物燃料の処理をより経済的に実現可能にするために重要である。
(トランスポータおよびβ−グルコシダーゼの相同分子種)
セロデキストリントランスポータに対するGenBankアクセション番号またはJoint Genome Institute(JGI)タンパク質ID(PID)番号は、Tuber melanosporum、CAZ81962.1;Pichia stipitis、ABN65648.2;Laccaria bicolor、EDR07962;Aspergillus oryzae、BAE58341.1;Phanerochaete chrysosporium、PID136620(JGI)(Martinez et al., 2004);Postia placenta、PID115604(JGI)(Martinez et al., 2009)である。Saccharomyces cerevisiaeのHXT1およびKluyveromyces lactisのLACPに対するGenBankアクセション番号は、それぞれDAA06789.1およびCAA30053.1である。P. chrysosporiumおよびP. placentaのゲノムはそれぞれ、genome.jgi-psf.org/Phchr1/Phchr1.home.htmlおよびgenome.jgi-psf.org/Pospl1/Pospl1.home.htmlにおいて利用可能であり得る。
セロデキストリントランスポータに対するGenBankアクセション番号またはJoint Genome Institute(JGI)タンパク質ID(PID)番号は、Tuber melanosporum、CAZ81962.1;Pichia stipitis、ABN65648.2;Laccaria bicolor、EDR07962;Aspergillus oryzae、BAE58341.1;Phanerochaete chrysosporium、PID136620(JGI)(Martinez et al., 2004);Postia placenta、PID115604(JGI)(Martinez et al., 2009)である。Saccharomyces cerevisiaeのHXT1およびKluyveromyces lactisのLACPに対するGenBankアクセション番号は、それぞれDAA06789.1およびCAA30053.1である。P. chrysosporiumおよびP. placentaのゲノムはそれぞれ、genome.jgi-psf.org/Phchr1/Phchr1.home.htmlおよびgenome.jgi-psf.org/Pospl1/Pospl1.home.htmlにおいて利用可能であり得る。
NCU00130の相同分子種であるセロデキストリンヒドロラーゼに対するGenBankアクセション番号は、T. melanosporum、CAZ82985.1;A. oryzae、BAE57671.1;P. placenta、EED81359.1;およびP. chrysosporium、BAE87009.1である。セロデキストリントランスポータの相同分子種を含んでいる他の生物は、細胞内β−グルコシダーゼであると推定されているGH3ファミリーにおける遺伝子を含んでおり(Bendtsen et al., 2004; Cantarel et al., 2009)、当該他の細胞は、Kluyveromyces lactis、CAG99696.1;Laccaria bicolor、EDR09330;Clavispora lusitaniae、EEQ37997.1;およびPichia stipitis、ABN67130.1である。
(株および培地)
本研究に使用された酵母株は、遺伝子型:MATa ura3-52 Lys2−801_amber ade2-101_ochre trp1−Δ63 his3−Δ200 leu2−Δ1を有しているYPH499であった(Sikorski et al., 1989)。100mg/Lのアデニンヘミサルフェートを補ったYPD培地において、上記酵母株を成長させた。100mg/Lのアデニンヘミサルフェートを補った適切な完全最少ドロップアウト(dropout)培地において形質転換された株(Becker et al., 2001)を成長させた。本研究に使用されたNeurospora crassaの株は、Fungal Genetics Stock Center(McCluskey 2004)から入手され、WT(FGSC2489)、およびセロビオーストランスポータの2つの欠失株(FGSC16575、ΔNCU00801.2およびFGSC17868、ΔNCU08114.2(Colot et al., 2006)を含んでいた。
本研究に使用された酵母株は、遺伝子型:MATa ura3-52 Lys2−801_amber ade2-101_ochre trp1−Δ63 his3−Δ200 leu2−Δ1を有しているYPH499であった(Sikorski et al., 1989)。100mg/Lのアデニンヘミサルフェートを補ったYPD培地において、上記酵母株を成長させた。100mg/Lのアデニンヘミサルフェートを補った適切な完全最少ドロップアウト(dropout)培地において形質転換された株(Becker et al., 2001)を成長させた。本研究に使用されたNeurospora crassaの株は、Fungal Genetics Stock Center(McCluskey 2004)から入手され、WT(FGSC2489)、およびセロビオーストランスポータの2つの欠失株(FGSC16575、ΔNCU00801.2およびFGSC17868、ΔNCU08114.2(Colot et al., 2006)を含んでいた。
(プラスミドおよびクローニング)
トランスポータを、2μプラスミドpRS426にクローン化した。pRS426は、改変されて、プライマー:ATATATGAGCTCGTGAGTAAGGAAAGAGTGAGGAACTATC(配列番号53)およびATATATACTAGTTGTTTTATATTTGTTGTAAAAAGTAGATAATTACTTCC(配列番号54)を用いて、SacIおよびSpeIの間に挿入されたS. cerevisiaeのPGK1プロモータを含んでいた。上述および後述のプライマーのすべてにおいて制限酵素部位には下線が付されている。それから、C末端にMycタグおよび最適化されたKozak配列(Miyasaka 1999)を有しているNCU00801を、プライマー:ATGGATCCAAAAATGTCGTCTCACGGCTCC(配列番号55)およびATGAATTCCTACAAATCTTCTTCAGAAATCAATTTTTGTTCAGCAACGATAGCTTCGGAC(配列番号56)を用いて、BamHIおよびEcoRIの間に挿入した。C末端にMycタグおよび最適化されたKozak配列を有しているNCU08114を、プライマー:ATACTAGTAAAAATGGGCATCTTCAACAAGAAGC(配列番号57)およびGCATATCGATCTACAAATCTTCTTCAGAAATCAATTTTTGTTCAGCAACAGACTTGCCCTCATG(配列番号58)を用いて、SpeIおよびClaIの間に挿入した。GFP融合体を作製するために、Gly−Ser−Gly−SerのN末端リンカーを有しているスパーフォルダーGFP(Pedelacq et al., 2006)を、プライマー:TATTAAATCGATGGTAGTGGTAGTGTGAGCAAGGGCGAGGAG(配列番号59)およびTATTAAGTCGACCTACTTGTACAGCTCGTCCATGCC(配列番号60)を用いて、PGK1プロモータを含んでいるpRS426プラスミドのClaIおよびSalIの間にまず挿入した。それから、トランスポータを以下のようにGFPに対して融合させた。プライマー:GCATGGATCCATGTCGTCTCACGGCTCC(配列番号61)およびTATAATGAATTCAGCAACGATAGCTTCGGAC(配列番号62)を用いて、NCU00801をBamHIおよびEcoRIの間に挿入した。プライマー:TATTAAACTAGTATGGGCATCTTCAACAAGAAGC(配列番号63)およびTTATAAGAATTCAGCAACAGACTTGCCCTCATG(配列番号64)を用いて、NCU08114をSpeIおよびEcoRIの間に挿入した。
トランスポータを、2μプラスミドpRS426にクローン化した。pRS426は、改変されて、プライマー:ATATATGAGCTCGTGAGTAAGGAAAGAGTGAGGAACTATC(配列番号53)およびATATATACTAGTTGTTTTATATTTGTTGTAAAAAGTAGATAATTACTTCC(配列番号54)を用いて、SacIおよびSpeIの間に挿入されたS. cerevisiaeのPGK1プロモータを含んでいた。上述および後述のプライマーのすべてにおいて制限酵素部位には下線が付されている。それから、C末端にMycタグおよび最適化されたKozak配列(Miyasaka 1999)を有しているNCU00801を、プライマー:ATGGATCCAAAAATGTCGTCTCACGGCTCC(配列番号55)およびATGAATTCCTACAAATCTTCTTCAGAAATCAATTTTTGTTCAGCAACGATAGCTTCGGAC(配列番号56)を用いて、BamHIおよびEcoRIの間に挿入した。C末端にMycタグおよび最適化されたKozak配列を有しているNCU08114を、プライマー:ATACTAGTAAAAATGGGCATCTTCAACAAGAAGC(配列番号57)およびGCATATCGATCTACAAATCTTCTTCAGAAATCAATTTTTGTTCAGCAACAGACTTGCCCTCATG(配列番号58)を用いて、SpeIおよびClaIの間に挿入した。GFP融合体を作製するために、Gly−Ser−Gly−SerのN末端リンカーを有しているスパーフォルダーGFP(Pedelacq et al., 2006)を、プライマー:TATTAAATCGATGGTAGTGGTAGTGTGAGCAAGGGCGAGGAG(配列番号59)およびTATTAAGTCGACCTACTTGTACAGCTCGTCCATGCC(配列番号60)を用いて、PGK1プロモータを含んでいるpRS426プラスミドのClaIおよびSalIの間にまず挿入した。それから、トランスポータを以下のようにGFPに対して融合させた。プライマー:GCATGGATCCATGTCGTCTCACGGCTCC(配列番号61)およびTATAATGAATTCAGCAACGATAGCTTCGGAC(配列番号62)を用いて、NCU00801をBamHIおよびEcoRIの間に挿入した。プライマー:TATTAAACTAGTATGGGCATCTTCAACAAGAAGC(配列番号63)およびTTATAAGAATTCAGCAACAGACTTGCCCTCATG(配列番号64)を用いて、NCU08114をSpeIおよびEcoRIの間に挿入した。
β−グルコシダーゼNCU00130を2μプラスミドpRS425にクローン化し、改変して、上述のPGK1プロモータを含めさせた。最適化されたKozak配列およびC末端の6×Hisタグを有しているNCU00130を、プライマーGCATACTAGTAAAAATGTCTCTTCCTAAGGATTTCCTCT(配列番号65)およびATACTGCAGTTAATGATGATGATGATGATGGTCCTTCTTGATCAAAGAGTCAAAG(配列番号66):を用いて、SpeIおよびPstIの間に挿入した。すべてのコンストラクトは、XhoIおよびKpnIの間にCyc転写ターミネータを含んでいた。すべてのN. crassa遺伝子をcDNAからPCRによって増幅させた。当該cDNAは、単独の炭素源として純粋なセルロース(Avicel)を有している最少培地において培養したN. crassa(FGSC2489)から単離されたmRNAから合成された。
(酵母成長アッセイ)
セロ−オリゴ糖に基づく成長を観察するために、適切なドロップアウトを有している5mLの完全な最少培地において、一晩にわたって改変した株を成長させた。25mLのddH2Oを用いて3回にわたって、これらの開始培養物を洗浄し、酵母窒素原礎培地(YNB)、適切な完全補助培地(Complete Supplemental Media)(CSM)ならびに0.1%(w/v)のセロビオース、または0.5%(w/v)のセロトリオースもしくはセロテトラオースにおいて0.1のOD(600nmにおける)まで再懸濁させた。0.4mLの最終のアッセイ容積および30℃において、最大振幅の一定速度において振とうさせながら、Bioscreen C(商標)においてアッセイを実施した。600nmにおいてか、または450〜580nmの広帯域のフィルタを用いて、ODの変化を測定した。成長速度を、成長曲線の直線部分から取り、独立した3つの実験の平均±これらの実験の間の標準偏差として記録した。セロビオースおよびセロトリオースを、Seikagaku Biobusiness Corporation(Tokyo, Japan)から入手した。
セロ−オリゴ糖に基づく成長を観察するために、適切なドロップアウトを有している5mLの完全な最少培地において、一晩にわたって改変した株を成長させた。25mLのddH2Oを用いて3回にわたって、これらの開始培養物を洗浄し、酵母窒素原礎培地(YNB)、適切な完全補助培地(Complete Supplemental Media)(CSM)ならびに0.1%(w/v)のセロビオース、または0.5%(w/v)のセロトリオースもしくはセロテトラオースにおいて0.1のOD(600nmにおける)まで再懸濁させた。0.4mLの最終のアッセイ容積および30℃において、最大振幅の一定速度において振とうさせながら、Bioscreen C(商標)においてアッセイを実施した。600nmにおいてか、または450〜580nmの広帯域のフィルタを用いて、ODの変化を測定した。成長速度を、成長曲線の直線部分から取り、独立した3つの実験の平均±これらの実験の間の標準偏差として記録した。セロビオースおよびセロトリオースを、Seikagaku Biobusiness Corporation(Tokyo, Japan)から入手した。
(NCU00130の精製およびその活性のアッセイ)
cbt1およびNCU00130を発現しているS. cerevisiaeの1Lの培養物を、完全最少培地において2.0ODまで成長させた。細胞を、遠心分離によって回収し、30mLの溶解緩衝液(50mMのNaH2PO4[pH8.0]、300mMのNaCl、10mMのイミダゾール、2mMのβ−ME、Complete(商標) Mini、EDTAを含んでいないプロテアーゼ阻害剤カクテル)に再懸濁させた。超音波によって細胞を溶解させ、溶解物を30分間にわたる15000gにおける遠心分離することによって不要物を取り除いた。溶解物を重力にしたがって流すことによって、ニッケル−NTA樹脂と結合させ、25mLの洗浄緩衝液(溶解緩衝液と同一であるが、20mMのイミダゾールを有している)を用いて3回にわたって洗浄した。5mLの溶出緩衝液(溶解緩衝液と同一であるが、150mMのイミダゾールを有している)を用いてNCU00130を溶出させ、適切な画分をプールし、保存緩衝液(リン酸緩衝生理食塩水(PBS))に交換し、等量に分注し、液体窒素において凍結させ、−80℃において保存した。SDS−PAGEによって純度を決定し(図22)、108750M−1cm−1の減衰係数を用いて280nmの吸光度からタンパク質濃度を決定した。
cbt1およびNCU00130を発現しているS. cerevisiaeの1Lの培養物を、完全最少培地において2.0ODまで成長させた。細胞を、遠心分離によって回収し、30mLの溶解緩衝液(50mMのNaH2PO4[pH8.0]、300mMのNaCl、10mMのイミダゾール、2mMのβ−ME、Complete(商標) Mini、EDTAを含んでいないプロテアーゼ阻害剤カクテル)に再懸濁させた。超音波によって細胞を溶解させ、溶解物を30分間にわたる15000gにおける遠心分離することによって不要物を取り除いた。溶解物を重力にしたがって流すことによって、ニッケル−NTA樹脂と結合させ、25mLの洗浄緩衝液(溶解緩衝液と同一であるが、20mMのイミダゾールを有している)を用いて3回にわたって洗浄した。5mLの溶出緩衝液(溶解緩衝液と同一であるが、150mMのイミダゾールを有している)を用いてNCU00130を溶出させ、適切な画分をプールし、保存緩衝液(リン酸緩衝生理食塩水(PBS))に交換し、等量に分注し、液体窒素において凍結させ、−80℃において保存した。SDS−PAGEによって純度を決定し(図22)、108750M−1cm−1の減衰係数を用いて280nmの吸光度からタンパク質濃度を決定した。
異なるセロデキストリン基質を用いた加水分解活性に基づいて、精製されたNCU00130をアッセイした。150μLのPBSおよび3mMのDTTにおける500μMの各糖とともに、5pmolの酵素をインキュベートすることによって活性を測定した。100μLであった、40分にわたって30℃において処理した反応物を移し、400μLの0.1MのNaOHにおいて反応を停止させた。CarboPac PA200カラムを有しているDionex ICS-3000用いたイオンクロマトグラフィーによって結果物を分析した。
(トランスポータの相同分子種の系統学的な分析)
CBT1およびCBT2のアミノ酸配列をネットワーク上のデータベースから入手した。T-Coffee(Notredame et al., 2000)を用いて、複数の配列の整列化を行った。PhyML version 3.0(Guindon and Gascuel 2003)を用いて、100のBootstrapsによって最大尤度の系統発生を決定した。両方のプログラムには、Phylogeny.fr(ウェブページphylogeny.fr/)を介してアクセスした。FigTree v.1.2.1(ウェブページtree.bio.ed.ac.uk/)を用いて結果の系統樹を可視化した。
CBT1およびCBT2のアミノ酸配列をネットワーク上のデータベースから入手した。T-Coffee(Notredame et al., 2000)を用いて、複数の配列の整列化を行った。PhyML version 3.0(Guindon and Gascuel 2003)を用いて、100のBootstrapsによって最大尤度の系統発生を決定した。両方のプログラムには、Phylogeny.fr(ウェブページphylogeny.fr/)を介してアクセスした。FigTree v.1.2.1(ウェブページtree.bio.ed.ac.uk/)を用いて結果の系統樹を可視化した。
(発酵およびSSF)
発酵実験およびSSF実験において、NCU00130を発現しており、かつMyc−タグつきのcbt1を発現しているか、またはトランスポータを発現していない酵母の間において比較を行った。完全最少培地において一晩にわたって好気性条件においてこれらの株を成長させ、25mLの水を用いて3回にわたって洗浄し、適切なCSMおよび2%(w/v)のセロビオースもしくは3%(w/v)の純粋なセルロース(Avicel)を加えた50mLのYNBにおいて最終のODが2.0になるまで、密閉した血清フラスコにおいて再懸濁させた。また、SSF反応物は、β−グルコシダーゼを補うことなく、50Filter Paper Units/gセルロースのろ過滅菌したCelluclast(Sigma C2730)を含んでいた。振とうさせながら30℃において嫌気的に反応を行った。示されている時点において、1mLのサンプルを取り出し、0.2μmのシリンジフィルタに通してろ過した。Aminex HPX-87Hカラムを用いたHPLCおよび屈折率の検出によって、ろ過物におけるエタノール、グルコースおよびセロビオースの濃度を決定した。
発酵実験およびSSF実験において、NCU00130を発現しており、かつMyc−タグつきのcbt1を発現しているか、またはトランスポータを発現していない酵母の間において比較を行った。完全最少培地において一晩にわたって好気性条件においてこれらの株を成長させ、25mLの水を用いて3回にわたって洗浄し、適切なCSMおよび2%(w/v)のセロビオースもしくは3%(w/v)の純粋なセルロース(Avicel)を加えた50mLのYNBにおいて最終のODが2.0になるまで、密閉した血清フラスコにおいて再懸濁させた。また、SSF反応物は、β−グルコシダーゼを補うことなく、50Filter Paper Units/gセルロースのろ過滅菌したCelluclast(Sigma C2730)を含んでいた。振とうさせながら30℃において嫌気的に反応を行った。示されている時点において、1mLのサンプルを取り出し、0.2μmのシリンジフィルタに通してろ過した。Aminex HPX-87Hカラムを用いたHPLCおよび屈折率の検出によって、ろ過物におけるエタノール、グルコースおよびセロビオースの濃度を決定した。
(N. crassaの成長およびAlamar Blue(登録商標)アッセイ)
WTのN. crassa(FGSC 2489)および同核状態のNCU08114(FGSC17868)(Colot et al., 2006)を、Fungal Genetics Research Center(McCluskey 2003)から入手し、バッフルなしの250mLのフラスコに入っている、2%のスクロースまたは純粋なセルロース(Avicel)を加えた50mLのVogel's塩および25℃において成長させた。16または28時間後にそれぞれ、100μLのAlamar Blue(登録商標)を加え、培養物を20分にわたって室温において培養した。このときに、1mLのサンプルを取り出し、細胞片をペレット化させ、Beckman Coulter Paradigmプレート読取り器において535/595nmの励起波長/放射波長を用いて、100μLの上清の蛍光発光を決定した。
WTのN. crassa(FGSC 2489)および同核状態のNCU08114(FGSC17868)(Colot et al., 2006)を、Fungal Genetics Research Center(McCluskey 2003)から入手し、バッフルなしの250mLのフラスコに入っている、2%のスクロースまたは純粋なセルロース(Avicel)を加えた50mLのVogel's塩および25℃において成長させた。16または28時間後にそれぞれ、100μLのAlamar Blue(登録商標)を加え、培養物を20分にわたって室温において培養した。このときに、1mLのサンプルを取り出し、細胞片をペレット化させ、Beckman Coulter Paradigmプレート読取り器において535/595nmの励起波長/放射波長を用いて、100μLの上清の蛍光発光を決定した。
(N. crassaのセロビオース輸送アッセイ)
WTのN. crassa(FGSC2489)、ならびにNCU00801(FGSC16575)およびNCU08114(FGSC17868)の同核状態の欠失株(Colot et al., 2006)を、Fungal Genetics Stock Center(McCluskey 2003)から入手し、106分生子/mLの播種物から始めて16時間にわたって、2%(w/v)のスクロースを加えた50mLのVogel's塩および25℃において、成長させた。遠心分離によって菌糸を回収し、Vogel's塩を用いて4回にわたって洗浄し、0.5%(w/v)の純粋なセルロース(Avicel)を加えたVogel's塩に移して、4時間にわたってトランスポータ発現を誘導させた。10mLの培養物を遠心分離によって回収し、Vogel's塩を用いて3回にわたって洗浄し、シクロヘキサン(100μg/mL)および90μMのそれぞれのセロデキストリン(セロビオース、セロトリオースまたはセロテトラオース)を加えた1mLのddH2Oに再懸濁させた。セロデキストリン消費を測定するために、15分後に100μLを取り出し、遠心分離によって不要物を除去し、900μLの0.1MのNaOHに移した。CarboPac PA200カラムを用いた、Dionex ICS-3000を有しているHPLCによって上清に残っている糖の量を決定した。電気化学検出器を用いてピークを検出した。
WTのN. crassa(FGSC2489)、ならびにNCU00801(FGSC16575)およびNCU08114(FGSC17868)の同核状態の欠失株(Colot et al., 2006)を、Fungal Genetics Stock Center(McCluskey 2003)から入手し、106分生子/mLの播種物から始めて16時間にわたって、2%(w/v)のスクロースを加えた50mLのVogel's塩および25℃において、成長させた。遠心分離によって菌糸を回収し、Vogel's塩を用いて4回にわたって洗浄し、0.5%(w/v)の純粋なセルロース(Avicel)を加えたVogel's塩に移して、4時間にわたってトランスポータ発現を誘導させた。10mLの培養物を遠心分離によって回収し、Vogel's塩を用いて3回にわたって洗浄し、シクロヘキサン(100μg/mL)および90μMのそれぞれのセロデキストリン(セロビオース、セロトリオースまたはセロテトラオース)を加えた1mLのddH2Oに再懸濁させた。セロデキストリン消費を測定するために、15分後に100μLを取り出し、遠心分離によって不要物を除去し、900μLの0.1MのNaOHに移した。CarboPac PA200カラムを用いた、Dionex ICS-3000を有しているHPLCによって上清に残っている糖の量を決定した。電気化学検出器を用いてピークを検出した。
(GFP蛍光発光および共焦点蛍光顕微鏡研究)
485/535nmの励起波長/放射波長を用いたBeckman Coulter Paradigmプレート読取り器において、Bulk-cellのGFP蛍光発光の測定を行った。488nmのレーザをともなうYokogawa CSU-X1の回転板ヘッドに取りつけられたLeica SD6000顕微鏡に基づく開口数1.4の100倍の油浸対物レンズを用いた、0.8〜1.2のOD(600nm)を有している細胞の共焦点蛍光顕微鏡実験を実施し、Metamorphソフトウェアによって制御した。200nmの間隔の大きさでもってZ系列を記録し、ImageJを用いて分析した。
485/535nmの励起波長/放射波長を用いたBeckman Coulter Paradigmプレート読取り器において、Bulk-cellのGFP蛍光発光の測定を行った。488nmのレーザをともなうYokogawa CSU-X1の回転板ヘッドに取りつけられたLeica SD6000顕微鏡に基づく開口数1.4の100倍の油浸対物レンズを用いた、0.8〜1.2のOD(600nm)を有している細胞の共焦点蛍光顕微鏡実験を実施し、Metamorphソフトウェアによって制御した。200nmの間隔の大きさでもってZ系列を記録し、ImageJを用いて分析した。
([3H]セロビオース輸送アッセイおよび動態パラメータ)
オイルストップ法(Arendt et al., 2007)を改良して、輸送アッセイを実施した。GFPと融合されたcbt1またはcbt2のいずれかを発現している酵母株を、選択培地において1.5〜3.0のOD(600nm)まで成長させ、氷冷したアッセイ緩衝液(30mMのMES−NaOH[pH5.6]および50mMのエタノール)を用いて3回にわたって洗浄し、20のODまで再懸濁させた。輸送反応を開始させるために、50μLの細胞を、100μLのシリコンオイル(Sigma 85419)の上に層状化された50μLの[3H]セロビオースに加えた。17000gにおいて1分にわたってオイルを介して細胞を回転させることによって反応を停止させ、チューブをエタノール/ドライアイスにおいて凍結させ、チューブの底に入っている細胞のペレットを、0.5MのNaOHに取り去った。ペレットを一晩にわたって可溶化させ、5mLのUltima Goldシンチレーション液を加え、Tri-Carb 2900TRシンチレーションカウンターにおいてCPMを決定した。[3H]セロビオースは、Moravek Biochemicals, Inc.から購入され、4Ci/mmolの特定の活性および99%を超える純度を有していた。種々のセロビオース濃度に対する3分間の全体における直線的な[3H]セロビオース取込み率を測定することによって、動態パラメータを決定した。SigmaPlot(登録商標)における非線形回帰による比率対セロビオース濃度のプロットに対して2変数の単一の直角双曲線関数を適合させることによって、VmaxおよびKm値を決定した。輸送アッセイを始める直前に、OD20における100μLの細胞からのGFP蛍光発光を測定することによって、トランスポータの存在量における差異について、Vmax値を標準化させた。文字として記録した動態パラメータは、独立した3つの実験からの平均±標準偏差である。250μMのそれぞれの競合物の存在下における20秒間にわたる全体の50μMの[3H]セロビオースの輸送を測定することによって、比較アッセイを行った。
オイルストップ法(Arendt et al., 2007)を改良して、輸送アッセイを実施した。GFPと融合されたcbt1またはcbt2のいずれかを発現している酵母株を、選択培地において1.5〜3.0のOD(600nm)まで成長させ、氷冷したアッセイ緩衝液(30mMのMES−NaOH[pH5.6]および50mMのエタノール)を用いて3回にわたって洗浄し、20のODまで再懸濁させた。輸送反応を開始させるために、50μLの細胞を、100μLのシリコンオイル(Sigma 85419)の上に層状化された50μLの[3H]セロビオースに加えた。17000gにおいて1分にわたってオイルを介して細胞を回転させることによって反応を停止させ、チューブをエタノール/ドライアイスにおいて凍結させ、チューブの底に入っている細胞のペレットを、0.5MのNaOHに取り去った。ペレットを一晩にわたって可溶化させ、5mLのUltima Goldシンチレーション液を加え、Tri-Carb 2900TRシンチレーションカウンターにおいてCPMを決定した。[3H]セロビオースは、Moravek Biochemicals, Inc.から購入され、4Ci/mmolの特定の活性および99%を超える純度を有していた。種々のセロビオース濃度に対する3分間の全体における直線的な[3H]セロビオース取込み率を測定することによって、動態パラメータを決定した。SigmaPlot(登録商標)における非線形回帰による比率対セロビオース濃度のプロットに対して2変数の単一の直角双曲線関数を適合させることによって、VmaxおよびKm値を決定した。輸送アッセイを始める直前に、OD20における100μLの細胞からのGFP蛍光発光を測定することによって、トランスポータの存在量における差異について、Vmax値を標準化させた。文字として記録した動態パラメータは、独立した3つの実験からの平均±標準偏差である。250μMのそれぞれの競合物の存在下における20秒間にわたる全体の50μMの[3H]セロビオースの輸送を測定することによって、比較アッセイを行った。
(大規模な酵母成長)
異なる炭素源に基づく成長を観察するために、適切なドロップアウトを有している5mLの完全最少培地において改変株を成長させた。25mLのddH2Oを用いて3回にわたってこれらの開始培養物を洗浄し、完全補助培地(CSM)および2%(w/v)のセロビオースを加えた50mLの酵母窒素原礎培地において0.1OD(600nmにおける)まで再懸濁させた。200rpmにおいて攪拌しながら、30℃においてバッフルなしの250mLのフラスコにおいて培養物を成長させた。OD(600nmにおける)の変化を定期的に取り出すことによって観察した。
異なる炭素源に基づく成長を観察するために、適切なドロップアウトを有している5mLの完全最少培地において改変株を成長させた。25mLのddH2Oを用いて3回にわたってこれらの開始培養物を洗浄し、完全補助培地(CSM)および2%(w/v)のセロビオースを加えた50mLの酵母窒素原礎培地において0.1OD(600nmにおける)まで再懸濁させた。200rpmにおいて攪拌しながら、30℃においてバッフルなしの250mLのフラスコにおいて培養物を成長させた。OD(600nmにおける)の変化を定期的に取り出すことによって観察した。
〔実施例10:セロデキストリンのトランスポータ機能にとって重要な残基の同定〕
本実施例では、配列決定分析および変異生成研究を用いてセロデキストリントランスポータにおける保存されている残基および機能的に重要な残基を同定した。さらなるトランスポータをさらに同定した。
本実施例では、配列決定分析および変異生成研究を用いてセロデキストリントランスポータにおける保存されている残基および機能的に重要な残基を同定した。さらなるトランスポータをさらに同定した。
セロデキストリントランスポータの種々の変異体NCU00801(cbt1)またはNCU08114(cbt2)ならびに野生型のβ−グルコシダーゼNCU00130を発現している酵母株の成長速度を、単独の炭素源としてセロビオースを用いて成長させた。QuickChange(登録商標) II Site-directed Mutagenesis Kit(Stratagene, La Jolla, CA)を用いて製造者の指示にしたがって、NCU00801の96位およびNCU08114の96位の残基を個々に変異させた。2%のセロビオースを有している−ura −leuの100mg/Lの合成の限定培地において株を成長させた。培養を独立した2つのコロニーから開始した。
図26(a、b)に示されている結果が表している通り、W66、L73、Y74、N87、Y89、D90、Q104、F107、G113、F120、Y123、D139、G142、K144、M147、G150、Q169、F170、G173、R174、G178、G180、P189、Y191、E194、P198、R201、Y208、W235、R236、Q242、257PESPRF262(配列番号67)、Y279、G283、E296、D307、K308、W310、D312、R325、G336、Y345、N369、D385、F462、P468、E476、T480、またはG486に置換を有しているNCU00801を発現している変異株は、野生株と比べて、25%の成長不良を示した。
NCU08114に対するアラニンスキャニング実験は、以下の残基:L38、Y39、G54、D56、F73、G91、P100、D104、G107、R108、M118、R139、F144、Q150、P154、E159、P163、H165、R166、Y173、N174、W199、Q214、222PESP225(配列番号68)、Y244、H245、D249、E258、E268、Q302、W303、S304、N306、Y312、F359、L360、F402、Y403、S404、Y414、E417、P420、Y421、K426、N442、N446、P447、W459、K460、E482、T483、L488、E489、E490、D496、およびG497が機能的に重要であることを示した(図26b)。
特に、モチーフ73LYF75、257PESP260(配列番号69)および278KYH280(NCU00801の残基の番号付与)は、29%のアミノ酸配列同一性を有している両方のトランスポータ(NCU00801の残基257PESP260(配列番号69)およびNCU08114の残基222PESP225(配列番号68))において機能的に重要であると考えられた。トランスポータにおいて全体的に保存されている残基(図26b、cにおける斜字体)、または特にβ結合型のトランスポータにおいて保存されているいくつかの残基(二重下線)(例えば、D90(NCU00801)、D56(NCU08114)、L73(NCU00801)およびL38(NCU08114))は、トランスポータ機能にとって重要である(下線)と実験的に示された。また、変異生成実験の結果は、NCU00801/NCU08114の分岐群に保存されている残基(キャップ)(例えば、Q168(NCU00801)およびQ214(NCU08114))が機能的に重要であることを示唆している。さらに、本実験において機能的に重要であると決定された複数の残基(例えば、L73(NCU00801)およびL38(NCU08114))は、S. cerevisiaeの糖トランスポータ(Hxt1/Hxt3)に保存されていることがこれまでに示されている。
また、異なる生物に由来するN. crassaのセロデキストリントランスポータの相同分子種を試験した(図27)。代表的な相同分子種を、Genescriptによって合成し、Cup1プロモータを含んでいる発現ベクターpRS426にBamHIおよびHindIII部位を用いてクローン化した。細胞内β−グルコシダーゼ(NCU00130)をともなっている酵母株YPH499を、これらのコンストラクトを用いて形質転換した。単独の炭素源としてセロビオースが存在しているときのこれらの株の成長速度を測定することによって、トランスポータ活性を決定した。
代替的に、推定の相同分子種を含んでいる異なる真菌株を、セロデキストリンを補った富栄養培地において培養した。総RNAを単離し、cDNAに逆転写させた。ポリメラーゼ連鎖反応(PCR)を用いて、cDNAから直接に推定のトランスポータ遺伝子を増幅させた。しかし、制御機構および発現パターンが真菌の種におけるセロデキストリントランスポータについて未知であるため、推定のトランスポータをコードしているcDNAは、培養条件の変更にもかかわらず必ずしも入手可能ではなかった。この場合に、GenBankから得たcDNA配列にしたがってプライマーを設計し、鋳型としてゲノムDNAを用いてエキソンを増幅させるために当該プライマーを使用した。それから、重複伸長PCRを用いて、エキソンを全長遺伝子にした。HXT7プロモータおよびHXT7ターミネータを含んでいるpRS424シャトルベクターに、DNAアセンブラ法を用いて生じたPCR産物をクローン化した。形質転換体から単離された酵母のプラスミドを用いてE. coliDH5αを形質転換し、元のトランスポータ遺伝子を増幅させるために使用したプラスミドを用いて、単離されたE. coliのプラスミドを確認用のPCRによってまず検査した。全体のオープンリーディングフレームを塩基配列決定法に供して、プラスミドの正しい構築を確認した。P. stipitis由来の相同分子種LAC2、LAC3、HXT2.1およびHXT2.6において、1つ以上の選択的なコドン(alternative codons)はSerをLeuに置換する。酵母の相同性組換えを介したDNAアセンブラ法を用いて、クローニング操作のほとんどを行った。pRS424−HXT7−GFPプラスミドを推定のセロデキストリントランスポータのクローニングに使用した。このプラスミドにおいてHXT7プロモータ、両方の末端にEcoRI部位を用いてフランキングしたGFP遺伝子、およびHXT7ターミネータを、ClaIおよびBamHIによって直鎖化されているpRS424シャトルベクター(New England Biolabs)に組み込んだ。HXT7プロモータおよびHXT7ターミネータと同一の配列を共有しているDNA断片を用いてフランキングさせた推定のトランスポータのPCR産物を、EcoRI消化したpRS424−HXT7−GFPを用いてCEN.PK2−1Cに、標準的な酢酸リチウム法にしたがってコトランスフェクションさせた。2%のグルコースを補ったSC−Trpプレートに生じた形質転換体の混合物を播いて、形質転換体を回復させた。推定のセロデキストリントランスポータの相同分子種、およびNCU00130を発現している酵母を、単独の炭素源としてのセロビオースに基づく成長について試験した。
推定のセロデキストリントランスポータの一覧および研究から得られた結果を表14に示す。
ある場合において、クローン化した相同分子種の配列が正確であることを確認し、これらのクローンを発現している酵母はセロビオースを利用可能であった。したがって、これらのクローンPichia stipitis由来のLAC2およびAspergillus clavatus由来のXP_001268541.1は、機能的なセロビオーストランスポータであることを確認した。他のクローンにおける公開されている配列から得られた異なる配列(例えば、挿入、欠失などを有している配列)を有しているクローン化した相同分子種を、ふたたびクローニングし、ふたたび配列決定し、S. cerevisiaeにおいてそれらを発現させることおよび成長速度を観察することによってセロビオーストランスポータ活性について同様に試験した。
NCU00801、NCU08114およびこれらのトランスポータの機能的な相同分子種の整列化配列を図28に示す。図28aにおける整列化配列は推定のトランスポータおよび確認済のトランスポータの両方を含んでおり、図28bにおける整列化配列は確認済のセロデキストリントランスポータのみを含んでいる。さらに、図28cはNCU00801およびNCU08144の整列化配列を示している。2つのトランスポータは29%のアミノ酸配列同一性を共有している。
セロデキストリントランスポータの機能にとって重要なモチーフを、糖トランスポータの間における複数の整列化配列を視覚的に検出することによって同定した。推定のセロデキストリントランスポータの相同分子種と確認済のセロデキストリントランスポータとの間の、T-COFFEEにおいて生成された複数の整列化配列から、セロデキストリントランスポータに共通のモチーフを同定した。これらのモチーフがセロデキストリントランスポータにとって非常に特異であることを確かめるために、S. cerevisiaeのヘキソーストランスポータと、ヒトのグルコーストランスポータGlut1と、N. crassaの単糖トランスポータとの間の、T-COFFEEにおいて生成された複数の整列化配列から、それらの非存在を確認した。
同定されたモチーフを以下に示す。モチーフにおいて、NCU00801の機能にとって重要である認められた残基に下線を付した。NCU08114の機能にとって重要である残基に上付き文字の“†”を付した。両方のトランスポータの機能にとって重要である残基に上付き文字の“*”を付した。すべてのモチーフをPROSITE表記法を用いて表した。PROSITEモチーフの読み方の一例として、次のモチーフ:[AC]−x−V−x(4)−{ED}は、[AlaまたはCys]−任意−Val−任意−任意−任意−任意−{GluまたはAspを除いて任意}(配列番号11)を解釈される。
セロデキストリントランスポータ、類似のすべての糖トランスポータは、膜貫通型の12のα−ヘリックスを有している。セロデキストリントランスポータのN末端およびC末端の両方は細胞内にある。
膜貫通型のヘリックス1の前にある配列は顕著な特徴を有していなかった。
膜貫通型のヘリックス1は、モチーフ[L* IVM]−Y*−[FL]−x(13)−[YF]−D*(配列番号1)を含んでいた。
膜貫通型のヘリックス2は、モチーフ[YF]−x(2)−G†−x(5)−[PVF]−x(6)−[DQ]*(配列番号2)を含んでいた。
ループによって接続されている膜貫通型のヘリックス2および膜貫通型のヘリックス3は、モチーフG*−R†−[RK]*(配列番号3)を含んでいた。
膜貫通型のヘリックス4は顕著な特徴を有していなかった。
膜貫通型のヘリックス5は、モチーフR*−x(6)−[YF]*−N†(配列番号4)を含んでいた。
膜貫通型のヘリックス6は、モチーフW* R−[IVLA]−P−x(3)−Q(配列番号5)を含んでいた。
膜貫通型のヘリックス6および膜貫通型のヘリックス7の間にある配列は、モチーフP*−E*−S*−P*−R−x−L−x(8)−A−x(3)−L−x(2)−Y*−H†(配列番号6)を含んでいた。
膜貫通型のヘリックス7は、モチーフF†−[GST]−Q*−x−S†−G−N†−x−[LIV](配列番号7)を含んでいた。
膜貫通型のヘリックス8は顕著な特徴を有していなかった。
膜貫通型のヘリックス9は顕著な特徴を有していなかった。
膜貫通型のヘリックス10、膜貫通型のヘリックス11およびこれらの間にある配列は、モチーフL−x(3)−[YIV]†−x(2)−E*−x−L−x(4)−R−[GA]−K†−G(配列番号8)を含んでいた。
膜貫通型のヘリックス12は顕著な特徴を有していなかった。
膜貫通型のヘリックス12の後ろに続く配列は顕著な特徴を有していなかった。
膜貫通型のヘリックス1の前にある配列は顕著な特徴を有していなかった。
膜貫通型のヘリックス1は、モチーフ[L* IVM]−Y*−[FL]−x(13)−[YF]−D*(配列番号1)を含んでいた。
膜貫通型のヘリックス2は、モチーフ[YF]−x(2)−G†−x(5)−[PVF]−x(6)−[DQ]*(配列番号2)を含んでいた。
ループによって接続されている膜貫通型のヘリックス2および膜貫通型のヘリックス3は、モチーフG*−R†−[RK]*(配列番号3)を含んでいた。
膜貫通型のヘリックス4は顕著な特徴を有していなかった。
膜貫通型のヘリックス5は、モチーフR*−x(6)−[YF]*−N†(配列番号4)を含んでいた。
膜貫通型のヘリックス6は、モチーフW* R−[IVLA]−P−x(3)−Q(配列番号5)を含んでいた。
膜貫通型のヘリックス6および膜貫通型のヘリックス7の間にある配列は、モチーフP*−E*−S*−P*−R−x−L−x(8)−A−x(3)−L−x(2)−Y*−H†(配列番号6)を含んでいた。
膜貫通型のヘリックス7は、モチーフF†−[GST]−Q*−x−S†−G−N†−x−[LIV](配列番号7)を含んでいた。
膜貫通型のヘリックス8は顕著な特徴を有していなかった。
膜貫通型のヘリックス9は顕著な特徴を有していなかった。
膜貫通型のヘリックス10、膜貫通型のヘリックス11およびこれらの間にある配列は、モチーフL−x(3)−[YIV]†−x(2)−E*−x−L−x(4)−R−[GA]−K†−G(配列番号8)を含んでいた。
膜貫通型のヘリックス12は顕著な特徴を有していなかった。
膜貫通型のヘリックス12の後ろに続く配列は顕著な特徴を有していなかった。
zhanglab.ccmb.med.umich.edu/I-TASSER/におけるI-TASSERサーバ(Roy et al., 2010).を用いて、NCU00801およびNCU08114の一次アミノ酸配列から、NCU00801およびNCU08114の同一性モデルを生成した。また、モチーフのマッピングをPYMOL(ウェブページpymol.org/)において行った。セロデキストリントランスポータの際立ったモチーフを有しているNCU00801およびNCU08114の同一性モデルを図29(a、b)に示す。図29(c)はNCU00801およびNCU08114の二次構造を示している。
〔実施例11:Neurospora crassaおよびPichia stipitisに由来するペントース特異的な新規のトランスポータの、Saccharomyces cerevisiaeにおける性質決定〕
本実施例において、バイオインフォマティックスの手法が、N. crassaおよびP. stipitisにおけるペントース特異的な新規のトランスポータを同定するために採用された。
本実施例において、バイオインフォマティックスの手法が、N. crassaおよびP. stipitisにおけるペントース特異的な新規のトランスポータを同定するために採用された。
(ペントース特異的なトランスポータのゲノムマイニング(mining))
バイオインフォマティックス研究
D−キシロース特異的なトランスポータを見つけるために、C. intermedia由来のD−グルコース/D−キシロース共輸送体のGxslをコードしている遺伝子(Leandro et al., 2006)、およびP. stipitis由来の性質決定されていない推定のL−アラビノース−プロトン共輸送体Aut1をコードしている遺伝子(遺伝子座タグPICST_87108)は、効率的にキシロースを利用する2つの異なる種N. crassaおよびP. stipitisの配列決定されたゲノム(Galagan et al., 2003; Jeffries et al., 2007)に対する、BLAST検索(ウェブページncbi.nlm.nih.gov/)におけるプローブとして使用した。公知のD−グルコース輸送活性または糖の輸送以外の活性を有している任意のタンパク質を分析から排除した。25%の最小の配列同一性の切捨てを用いて、17の推定のペントーストランスポータ遺伝子を、P. stipitis由来のAUT1に加えて、同定した(表15)。これらの推定のペントーストランスポータ遺伝子はC. intermediaのGxslまたはP. stipitisのAut1のいずれかと25〜50%の同一性を共有していた。推定の17のペントーストランスポータのすべては、糖輸送タンパク質または未知の活性を有している未知のタンパク質のいずれかとの注釈が付されていた。また、P. stipitisに由来するD−グルコーストランスポータ遺伝子SUT1およびSUTを、比較のためにクローニングした。
バイオインフォマティックス研究
D−キシロース特異的なトランスポータを見つけるために、C. intermedia由来のD−グルコース/D−キシロース共輸送体のGxslをコードしている遺伝子(Leandro et al., 2006)、およびP. stipitis由来の性質決定されていない推定のL−アラビノース−プロトン共輸送体Aut1をコードしている遺伝子(遺伝子座タグPICST_87108)は、効率的にキシロースを利用する2つの異なる種N. crassaおよびP. stipitisの配列決定されたゲノム(Galagan et al., 2003; Jeffries et al., 2007)に対する、BLAST検索(ウェブページncbi.nlm.nih.gov/)におけるプローブとして使用した。公知のD−グルコース輸送活性または糖の輸送以外の活性を有している任意のタンパク質を分析から排除した。25%の最小の配列同一性の切捨てを用いて、17の推定のペントーストランスポータ遺伝子を、P. stipitis由来のAUT1に加えて、同定した(表15)。これらの推定のペントーストランスポータ遺伝子はC. intermediaのGxslまたはP. stipitisのAut1のいずれかと25〜50%の同一性を共有していた。推定の17のペントーストランスポータのすべては、糖輸送タンパク質または未知の活性を有している未知のタンパク質のいずれかとの注釈が付されていた。また、P. stipitisに由来するD−グルコーストランスポータ遺伝子SUT1およびSUTを、比較のためにクローニングした。
推定のペントーストランスポータのクローニング
D−キシロースまたはL−アラビノースを炭素源として補った富栄養培地において、N. crassaおよびP. stipitisを培養した。総RNAを単離し、cDNAに逆転写した。ポリメラーゼ連鎖反応(PCR)を使用して、推定のトランスポータ遺伝子をcDNAから直接に増幅させた。しかし、真菌の種における調節機構および発現パターンが未知であるため、上記推定のペントーストランスポータをコードしているcDNAは、培養条件の変更にもかかわらず、必ずしも入手可能ではなかった。これらの場合、GenBankから提供されている対応するcDNA配列にしたがってプライマーを設計し、使用して、ゲノムDNAを鋳型として用いてエキソンを増幅させた。酵母の相同性組換えを介したDNAアセンブラ構築法(Shao et al., 2009)を用いて、生じたPCR産物をpRS424−HXT7−GFPシャトルベクターにクローニングした。このプラスミドにおいて、HXT7プロモータ、両端にEcoRI部位をフランキングさせたGFP遺伝子、およびHXT7ターミネータを、ClaIおよびBamHIによって直鎖化したpRS424シャトルベクター(New England Biolabs)に組み込んだ。HXT7のプロモータおよびターミネータと同一の配列を共有しているDNA断片を用いてフランキングした推定のペントーストランスポータのPCR産物(図30a)を、EcoRIによって消化したpRS4224−HXT7−GFPとともにEuroscarf(Frankfurt, Germany)から購入したS. cerevisiaeのCEN.PK2−1C(MATα leu2−3、112 ura3−52、trp1−289、his3−ΔMAL2−8c)に、標準的な酢酸リチウム法を用いてコトランスフェクションした。2%のD−グルコースを補ったSC−Trpプレートに、生じた形質転換混合物を播いた。
D−キシロースまたはL−アラビノースを炭素源として補った富栄養培地において、N. crassaおよびP. stipitisを培養した。総RNAを単離し、cDNAに逆転写した。ポリメラーゼ連鎖反応(PCR)を使用して、推定のトランスポータ遺伝子をcDNAから直接に増幅させた。しかし、真菌の種における調節機構および発現パターンが未知であるため、上記推定のペントーストランスポータをコードしているcDNAは、培養条件の変更にもかかわらず、必ずしも入手可能ではなかった。これらの場合、GenBankから提供されている対応するcDNA配列にしたがってプライマーを設計し、使用して、ゲノムDNAを鋳型として用いてエキソンを増幅させた。酵母の相同性組換えを介したDNAアセンブラ構築法(Shao et al., 2009)を用いて、生じたPCR産物をpRS424−HXT7−GFPシャトルベクターにクローニングした。このプラスミドにおいて、HXT7プロモータ、両端にEcoRI部位をフランキングさせたGFP遺伝子、およびHXT7ターミネータを、ClaIおよびBamHIによって直鎖化したpRS424シャトルベクター(New England Biolabs)に組み込んだ。HXT7のプロモータおよびターミネータと同一の配列を共有しているDNA断片を用いてフランキングした推定のペントーストランスポータのPCR産物(図30a)を、EcoRIによって消化したpRS4224−HXT7−GFPとともにEuroscarf(Frankfurt, Germany)から購入したS. cerevisiaeのCEN.PK2−1C(MATα leu2−3、112 ura3−52、trp1−289、his3−ΔMAL2−8c)に、標準的な酢酸リチウム法を用いてコトランスフェクションした。2%のD−グルコースを補ったSC−Trpプレートに、生じた形質転換混合物を播いた。
Zymoprep Yeast Plasmid Miniprep II((Zymo Research, Orange, CA)を用いて形質転換体から単離した酵母のプラスミドを、Escherichia coliのDH5a細胞(Cell Media Facility, Urbana-Champaignにあるイリノイ大学, Urbana, IL)に再びトランスフェクションした。QIAprep Spin Miniprep Kit(QIAGEN, Valencia, CA)を用いて、上記プラスミドを単離し、それから元のトランスポータ遺伝子の増幅に使用したプライマーを用いた確認的なPCRによって確認した。また、オープンリーディングフレームの全体をDNA配列決定に供して、正しい構成を確認した(Core Sequencing Facility, University of Illinois at Urbana-Champaign, Urbana, IL)。DNA配列決定の結果を、Sequencher 4.7(Gene Codes Corporation, Ann Arbor, MI)を用いて、データベースにおける配列と比較した。クローン化された推定のトランスポータのすべての配列は配列番号33〜52に記載されている。
合成のドロップアウト培地(アミノ酸および硫酸アンモニウムなしの0.17%のDifco yeast nitrogen base、0.5%の硫酸アンモニウム、0.05%のアミノ酸のドロップアウト混合物)において酵母株を培養して、プラスミドを保持させた。2%の糖を補ったYPA培地(1%の酵母抽出物、2%のペプトン、0.01%のアデニンヘミサルフェートスルフェート)を使用して、プラスミドを有していない酵母株を成長させた。S. cerevisiae株を、好気性成長のために30℃および250rpmにおいて培養し、酸素制限条件のために30℃および100rpmにおいて培養した。特に説明がない限り、細胞操作のために好気性条件下において酵母株を成長させた。37℃、250rpmおよびLuriaブロス(Fisher Scientific, Pittsburg, PA)においてE. coli株を培養した。すべての制限酵素をNew England Biolabs(Ipswich, MA)から購入した。すべての化学薬品をSigma Aldrich(St. Louis, MO)またはFisher Scientificから購入した。
(クローン化した推定のトランスポータのトランスポータ活性アッセイ)
ペントース糖の細胞内蓄積
クローン化した推定のペントーストランスポータを、S. cerevisiaeの糖トランスポータ欠失株において過剰発現させ、ペントース糖の取込みが測定された。推定のペントーストランスポータのD−キシロース取込み能を、細胞内のD−キシロースおよびキシリトールの濃度の総和によって決定した。S. cerevisiae内に蓄積したD−キシロースは、内因性のアルドースリダクターゼの存在のために、キシリトールへと部分的に変換され得る。D−キシロースおよびキシリトールの両方を、浸透圧を利用して抽出し、高速液体クロマトグラフィー(HPLC)を用いて分析した。
ペントース糖の細胞内蓄積
クローン化した推定のペントーストランスポータを、S. cerevisiaeの糖トランスポータ欠失株において過剰発現させ、ペントース糖の取込みが測定された。推定のペントーストランスポータのD−キシロース取込み能を、細胞内のD−キシロースおよびキシリトールの濃度の総和によって決定した。S. cerevisiae内に蓄積したD−キシロースは、内因性のアルドースリダクターゼの存在のために、キシリトールへと部分的に変換され得る。D−キシロースおよびキシリトールの両方を、浸透圧を利用して抽出し、高速液体クロマトグラフィー(HPLC)を用いて分析した。
E. Boles教授の研究室(Institut fur Mikrobiologie, Heinrich-Heine-Universitat, Universitatsstr. 1, Geb. 26.12.01, D-40225 Dusseldorf, Germany)から贈られた糖トランスポータのノックアウトS. cerevisiae株のEBY.VW4000(CEN.PK2−1c Δhxt1−17、Δstl1、Δagt1、Δydl247w、Δyjr160c、Δgal2)は、20を超える糖のトランスポータおよびセンサ(HXT1-17およびGAL2が挙げられる)の同時のノックアウトを有していた。単独の炭素源としてのD−グルコースに基づく成長は、この株において消失しており、異なる糖の輸送系を介したマルトースの取込みが保持されていた。また、EBY.VW4000株は、HPLCアッセイの条件下において最小のペントース取込みを示し、組換え体のD−キシロース取込みを試験するために好適な宿主になった。標準的な酢酸リチウム法を用いて、クローン化された推定のペントーストランスポータを過剰発現するプラスミドをEBY.VW4000株にトランスフェクションし、糖の取込み活性を測定するために使用した。
2%のマルトースを補った2mLのSC-Trp培地において細胞をまず培養した。それから、種菌培養物を使用して、250mLのフラスコにおける50mLの培養物に播種した。成長の24時間後に遠心分離によって細胞を回収し、2%のD−キシロースまたはL−アラビノースを補ったYPA培地に再懸濁させて、最終的なOD600を10にした。30分間、60分間、120分間、および24時間における5mLの培養物を細胞内の糖の濃度を測定するために採取した。氷冷した水を用いて培養物のサンプルを洗浄し、3mLの脱イオン化水に再懸濁した。250rpmにおいて攪拌しながら2日にわたって37℃において、細胞の懸濁物をインキュベートした。HPLC分析の前に0.22μmのPESフィルタ(Corning, Lowell, MA)に、生じた細胞の懸濁物を通してろ過した。BioRad HPX-87Cカラム(BioRad Laboratories, Hercules, CA)および低温−蒸発光散乱検出器Shimadzu ELSD-LTII(Shimadzu)を備えているShimadzu HPLCを用いて、糖および対応する糖アルコールの濃度(後述されている)の濃度を、製造者のプロトコルにしたがって測定した。OD10未満における細胞培養物の1mLについて浸透圧を介して抽出された糖のmg数として、糖の取込み活性を算出した。
種々の推定のペントーストランスポータは、D−グルコース、D−キシロースまたは両方の取込みにおいて活性を示すことが同定された。D−グルコースは酵母内においていったん代謝され得るので、D−グルコース輸送活性は、細胞内のD−グルコース濃度を測定することによって決定され得ない。しかし、通常、EBY.VW4000株は、単独の炭素源としてD−グルコースを含んでいる培地において成長できないので、推定のペントーストランスポータを用いて形質転換された株の、D−グルコースに基づく成長は、推定のトランスポータがD−グルコース輸送活性を有していることを示した。
SUT3(Xyp37)、XUT3(Xyr33)、SUT2(Ap31)、NCU04963(An29−2)およびNCU06138(Xy31)は、D−グルコースに基づくEBY.VW4000株の成長を回復させ、したがってグルコース輸送活性を発揮させた。また、SUT3、XUT3、SUT2およびNCU04963は、キシロース輸送活性を有しており、NCU04963およびNCU06138はアラビノース輸送活性を示した(図3)。残りの推定のトランスポータはD−グルコースに基づく成長を可能にすることができず、またそれらのほとんどは任意のペントース輸送活性を示さなかった。しかし、NCU00821およびSTL12/XUT6はキシロース輸送活性を示し、XUT1はアラビノース輸送活性を示し、それらがペントースに特異的な糖トランスポータであり得ることを示していた(図32)。
P. stipitisに由来するSTL12/XUT6およびXUT1がD−グルコース輸送活性なしで、実際にペントース特異的なトランスポータであることをさらに確認するために、14C標識したD−グルコース、D−キシロースおよびL−アラビノースを基質として用いて、糖取込みアッセイを実施した。STL12/XUT6およびNCU00821を過剰発現しているEBY.VW4000株のD−グルコース取込み活性およびL−アラビノース取込み活性は、両方のトランスポータのD−キシロース取込み動態を決定するために使用されたアッセイの条件下において測定されるには低すぎることが分かった。
14C標識したD−グルコース、D−キシロースおよびL−アラビノースを、90%のエタノールにおける溶液としてAmerican Radiolabeled Chemicals(St. Louis, MO)から購入した。放射線標識した糖を、ドラフトにおいてまず乾燥させ、それから水に再懸濁させた。約40000dpm/μLの固有の放射能を有している1.33Mおよび1Mの濃度における糖の溶液、ならびに約20000dpm/μLの固有の放射能を有している500mM、350mM、250mM、100mMおよび50mMの濃度における糖の溶液を糖の取込みアッセイに使用した。対数期における細胞の培養物を、回収し、氷冷した水を用いて2回にわたって洗浄し、pH5における100mMのTris−クエン酸緩衝液の1mLにつき約60mgの乾燥細胞重量(DCW)まで再懸濁させた。160μLの細胞懸濁物の3つの分取物を24時間にわたって65℃において乾燥させて、DCWを決定した。残りの細胞懸濁物を使用前まで氷上に静置しておいた。糖の取込みアッセイのために、細胞懸濁物を、40μLの放射性標識した糖の溶液と40または60秒にわたって(正確に時間を測定した)混合した。反応を、シリンジによって10mLの氷冷した水を加えることによって停止させた。0時点のサンプルを、放射性標識した溶液の入っている培養チューブに対して、氷冷した水および細胞懸濁物を同時に加えることによって入手した。それから、混合物を、40%の糖溶液にあらかじめ浸しておいたWhatman GF/Cフィルタ(Whatman, Florham Park, NJ)に通して直ちにろ過し、氷冷した15mLの水を用いて洗浄した。ろ過物を、3mLのEcono Iシンチレーションカクテル(Fisher Scientific)に入れ、Beckman LS6500シンチレーションカウンター(Beckman Coulter, Brea, CA)を用いて1分にわたってカウントした。すべての時点のデータは3つの独立した実験において測定された。輸送された糖のmmol数/時間/乾燥細胞重量の1グラムとして、糖の取込みを算出した。
また、STL12/XUT6、NCU00821またはXUT1を過剰発現しているEBY.VW4000株におけるD−キシロースおよびL−アラビノースの細胞内における蓄積を、HPLCを用いて測定した。30分間、60分間、120分間および24時間にわたってペントース糖とともにインキュベートした細胞培養物をHPLCによって分析した。24時間のインキュベーションの後に、STL12/XUT6またはNCU00821を過剰発現しているEBY.VW4000株は、D−キシロース取込み活性を示し、XUT1を過剰発現しているEBY.VW4000株は、L−アラビノース取込み活性を示した(図33)。
細胞内における糖の蓄積のHPLC分析をともなった、14C標識した糖の取込みアッセイによって、リグノセルロース性の加水分解物における最も豊富な3つの単糖(D−グルコース、D−キシロースおよびL−アラビノース)のなかでも、STL12/XUT6およびNCU00821はD−キシロースの取込みを担っており、XUT1はL−アラビノースの取込みを担っていることが確認された。注目すべき点は、D−キシロースの取込みについて最も研究されている糖トランスポータは、D−キシロースに対してより、D−グルコースに対して高い取込み活性を有している。適応性の進化の後にTrichoderma reesei由来のTrxlt1のみは、D−キシロース特異的な取込み活性を示した(Saloheimo et al., 2007)。このデータによって、P. stipitis由来のSTL12/XUT6およびN. crassa由来のNCU00821は、S. cerevisiaeに導入され、かつ実験的に初めて確認された天然に存在しているD−キシロース特異的なトランスポータであることが示された。同様に、P. stipitis由来のXUT1はS. cerevisiaeに導入され、かつ実験的に初めて確認された天然に存在しているL−アラビノース特異的なトランスポータであることが示された。
動態パラメータ
14C標識した糖の取込みアッセイを用いて、NCU00921、STL12/XUTR6およびXUT1を介したD−キシロース輸送の動態パラメータを決定した。アッセイ条件下における糖の取込みは、始めの60秒間において直線的な連なりであった(図34)。NCU00821、STL12/XUTR6またはXUT1を過剰発現しているEBY.VW4000株を、標識したD−キシロースまたはL−アラビノースとともに、40秒間または60秒にわたってインキュベートした後に、氷冷した水を加えて、さらなる糖の取込みを停止させた。それから、液体シンチレーションカウンターを用いた測定の前に反応混合物を、ろ過し、洗浄した。Originソフトウェア(OriginLab Corporation, Northampton, MA)を用いた非線形回帰によって糖の取込み率および基質の濃度を、ミカエリス−メンテンの方程式に適合させた。NCU00821またはSTL12/XUT6のみを有しているEBY.VW4000株によるD−キシロース取込みに関するKm値は、それぞれ175.7±21.4および56.0±9.4mMであった。対応するVmax値はそれぞれ36.7±2.8および41.5±2.3μmol/DCWの1gであった。同様に、XUT1を有しているEBY.VW4000株によるD−キシロース取込みに関するKm値およびVmax値は、それぞれ48.0±13.2mMおよび5.6±1.6μmol/DCWの1gであった。
14C標識した糖の取込みアッセイを用いて、NCU00921、STL12/XUTR6およびXUT1を介したD−キシロース輸送の動態パラメータを決定した。アッセイ条件下における糖の取込みは、始めの60秒間において直線的な連なりであった(図34)。NCU00821、STL12/XUTR6またはXUT1を過剰発現しているEBY.VW4000株を、標識したD−キシロースまたはL−アラビノースとともに、40秒間または60秒にわたってインキュベートした後に、氷冷した水を加えて、さらなる糖の取込みを停止させた。それから、液体シンチレーションカウンターを用いた測定の前に反応混合物を、ろ過し、洗浄した。Originソフトウェア(OriginLab Corporation, Northampton, MA)を用いた非線形回帰によって糖の取込み率および基質の濃度を、ミカエリス−メンテンの方程式に適合させた。NCU00821またはSTL12/XUT6のみを有しているEBY.VW4000株によるD−キシロース取込みに関するKm値は、それぞれ175.7±21.4および56.0±9.4mMであった。対応するVmax値はそれぞれ36.7±2.8および41.5±2.3μmol/DCWの1gであった。同様に、XUT1を有しているEBY.VW4000株によるD−キシロース取込みに関するKm値およびVmax値は、それぞれ48.0±13.2mMおよび5.6±1.6μmol/DCWの1gであった。
D−キシロースを消化する天然に存在する真菌の種において、高い親和性のD−キシロース−プロトンの共輸送系および低い親和性のD−キシロース促進性の拡散系の両方が存在している。これらの2つの系のKm値は、上記共輸送系について0.4〜4mMであり、促進性の拡散系について約140mMであると決定された(Leandro et al., 2006; Stambuk et al., 2003)。これらの値はS. cerevisiaeにおけるD−グルコース取込みの親和性に近く、D−グルコース取込みは高い親和性の系について1.5mMのKmを有しており、低い親和性の系について20mMのKmを有している(Lang and Cirillo 1987; Ramos et al., 1988)。残念ながら、野生型のS. cerevisiaeのD−キシロース取込みの親和性は、D−キシロースに対する親和性より2桁低い大きさである。S. cerevisiaeにおけるD−キシロース取込みについてKm値は、高い親和性の系についてわずか190mMであり、低い親和性の系についてわずか1.5mMである(Kotter and Ciriacy, 1993)。新たに見出されたD−キシロース特異的なトランスポータの親和性は、天然に存在するD−キシロースを消化する酵母における高い親和性のD−キシロース取込みの系と比べて低かった。しかし、野生型のS. cerevisiaeと比べて、NCU00821およびSTL12/XUT6はD−キシロースに対してより高い親和性を示した。特にSTL12/XUT6およびXUT1によるD−グルコース取込みのKmは、野生型のS. cerevisiaeにおけるトランスポータによるキシロース取込みのKmのわずか1/4であった。また、D−キシロース特異的なトランスポータのKm値は、Gxf1(88mMのKm)およびSut1(145mMのKm)のKm値と近く、組換えS. cerevisiaeにおけるD−キシロース発酵を向上させていることを示している(Runquist et al., 2009; Katahira et al., 2008)。したがってD−キシロース発酵は、新たに見出されたD−キシロース特異的なこれらのトランスポータをS. cerevisiaeに導入することによって向上され得る。
(糖トランスポータの細胞内の局在化)
糖トランスポータは膜貫通型のタンパク質であり、細胞膜における正しいフォールディングおよび局在化はそれらが機能的であるために必要である。推定のペントーストランスポータがクローン化されたときにシグナルペプチドを特に加えていないので、D−キシロース特異的なトランスポータが細胞膜に正しく局在化していることを確認することが重要であった。これは、S. cerevisiaeと比べて非常に異なる生理機能を示す糸状菌N. crassaからクローン化されたNCU00821のような推定のペントーストランスポータに対して特にあてはまる。S. cerevisiaeにおけるD−キシロース特異的なトランスポータの細胞への局在化を調べるために、NCU00821、STL12/XUT6およびXUT1のC末端にリンカーを介して緑色蛍光タンパク質(GFP)を融合させ、それらの局在化を蛍光発光イメージングによって観察した。
糖トランスポータは膜貫通型のタンパク質であり、細胞膜における正しいフォールディングおよび局在化はそれらが機能的であるために必要である。推定のペントーストランスポータがクローン化されたときにシグナルペプチドを特に加えていないので、D−キシロース特異的なトランスポータが細胞膜に正しく局在化していることを確認することが重要であった。これは、S. cerevisiaeと比べて非常に異なる生理機能を示す糸状菌N. crassaからクローン化されたNCU00821のような推定のペントーストランスポータに対して特にあてはまる。S. cerevisiaeにおけるD−キシロース特異的なトランスポータの細胞への局在化を調べるために、NCU00821、STL12/XUT6およびXUT1のC末端にリンカーを介して緑色蛍光タンパク質(GFP)を融合させ、それらの局在化を蛍光発光イメージングによって観察した。
C末端にGFPを有しているペントース特異的なトランスポータの融合タンパク質をトランスポータ局在化研究のために構築した。GSリンカー(Gly−Gly−Gly−Gly−Ser−Gly−Gly−Gly−Gly−Ser(配列番号70))を、トランスポータおよびGFPの間に挿入した。PCRプライマーによってGFPのオープンリーディングフレームのN末端にGSリンカーを付加させ、5’末端におけるトランスポータと相同なヌクレオチド配列および3’末端におけるHXT7ターミネータを用いてフランキングされているGSリンカー−GFPのPCR産物を生じた。元のpRS424−HXT7トランスポータのコンストラクトからトランスポータ遺伝子を増幅させて、5’末端におけるHXT7プロモータおよび3’末端におけるGSリンカー−GFPと同一のヌクレオチド配列を用いてフランキングされたトランスポータのDNA断片を生成させた。それから、EcoRIを用いて消化したpRS424−HXT7−GFPとともに、これらの2つの断片をS. cerevisiae株のCEN.PK2−1Cにコトランスフェクションした。生じた形質転換混合物を2%のD−グルコースを補ったSC−Trpプレートに播いた。
2%のマルトースを補った2mLのSC−Trp液体培地に単一のコロニーを播種した。細胞培養物を対数期に回収した。遠心分離チューブにおいて、10μLのHoechst 33342核染料(Invitrogen, Carlsbad, CA)を用いて10分にわたって室温において、250μLの細胞培養物を染色した。それから、細胞培養物の小さな液滴を一片のカバーガラス上に移し、Andor Technology Revolution System Spinning Disk Confocal Microscope(Core facilities, Institute for Genomic Biology, University of Illinois at Urbana-Champaign, Urbana, IL)を用いて、蛍光画像を撮影した。画像分析および可視化のソフトウェアImaris(Bitplane, Saint Paul, MN)を用いて、画像を処理した。
ペントース特異的なトランスポータを過剰発現している酵母株は、細胞の周辺領域に特有の蛍光発光の円光を示した(図35)。NCU00821およびXUT1について、ほとんどすべてのGFP蛍光発光は細胞膜に現れ、STL12/XUT6を過剰発現している細胞における蛍光発光の大部分は、細胞質に留まっていた。これは、膜タンパク質の上昇した発現に起因するSTL12/XUT6の非効率的な輸送を示し得る。また、すべての細胞が蛍光発光を示し、トランスポータの発現は最適ではなかったことを示していたことに注意されたい。発現レベルを変更すること、および/または組換えS. cerevisiaeのゲノムにトランスポータ遺伝子を組み込むことによって、トランスポータの発現のさらなる向上を実現し得る。
(ペントーストランスポータの種類の決定)
S. cerevisiaeには2種類の糖トランスポータ、共輸送体およびファシリテータ(facilitators)がある。通常、糖の共輸送体は糖に対する高い親和性を示す。一方で、ファシリテータを介した糖の取込みはプロトン輸送と連動しておらず、通常、ファシリテータは糖取込みの低い親和性を示す(Leandro et al., 2006)。EBY.VW4000株において発現されているNCU00821、STL12/XUT6およびXUT1について、共輸送体アッセイを実施した。
S. cerevisiaeには2種類の糖トランスポータ、共輸送体およびファシリテータ(facilitators)がある。通常、糖の共輸送体は糖に対する高い親和性を示す。一方で、ファシリテータを介した糖の取込みはプロトン輸送と連動しておらず、通常、ファシリテータは糖取込みの低い親和性を示す(Leandro et al., 2006)。EBY.VW4000株において発現されているNCU00821、STL12/XUT6およびXUT1について、共輸送体アッセイを実施した。
トランスポータの種類を決定するために、USB通信モジュールおよびDirect pHソフトウェアを備えているSeven MultiのpHメータ(Mettler Toledo, Columbus, OH)を用いて、D−キシロース、L−アラビノースまたはD−グルコースを含んでいる非緩衝化の細胞懸濁液において、ペントース特異的なトランスポータを過剰発現しているEBY.VW4000株のpH変化を測定した。ペントース特異的なトランスポータをコードしているプラスミドをEBY.VW4000株に入れ、2%のマルトースを補ったSC−Trp培地に播いた。2%のマルトースを補ったSC−Trp培地に単一のコロニーを播種した。それから種培養物を用いて2Lのフラスコに入っている400mLの培養物に播種した。OD1以下の培養物を回収し、氷冷した水を用いて2回にわたって洗浄した。細胞のペレットを4mLの水に再懸濁させ、使用するまで氷上に静置した。共輸送体アッセイのために、50mLの容量の水ジャケットつきの(water-jacketed)ビーカーにpH電極を挿入した。当該ビーカーは、25℃に維持されており、電磁攪拌機を備えている。25℃に保たれている23mLの脱イオン水および1mLの細胞懸濁物をビーカーに加えた。pHを5に調節し、基底線を得た。pH5を有している1mLの50%の糖溶液を加えながら、pH変化を記録した。
図36には、マルトースを加えた後における非緩衝化の細胞懸濁物におけるpH変化が示されている。報告されていた通り、非緩衝化のS. cerevisiae細胞の懸濁物におけるpHはマルトースの添加にしたがって上昇した。非緩衝化の細胞懸濁物に1mLの50%のマルトースを加えて、pH記録システムが機能していることを確認した。サンプルにおいて観察されたpH上昇は、pH記録計が実験環境における一過性のpH変化を観察可能であることを示していた。
非緩衝化の細胞懸濁物におけるpHの上昇は、ペントース特異的なトランスポータのいずれについても観察されず、これらのトランスポータを介したペントース取込みはプロトン輸送と関連していないことが示された(図37)。したがって、NCU00821、STL12/XUT6およびXUT1はペントースのファシリテータであると決定された。
この結果は、NCU00821およびSTL12/XUT6の動態パラメータが、D−キシロースを消化する天然に存在する酵母におけるD−キシロースの促進性の低い親和性の拡散系の動態パラメータと類似しているという事実と一致していた。共輸送体がD−キシロースに対するより高い親和性を有しているという事実にもかかわらず、共輸送体の過剰発現は、プロトンの勾配を作り出すためのATP要求に起因して、D−キシロースを消化する株による糖の利用を常に促進する訳ではない。実際に、D−キシロース発酵に有益であると示されているトランスポータのほとんどは、ファシリテータである(Runquist et al., 2009; Katahira et al., 2008)。
(p−キシロース特異的なトランスポータの異種性の過剰発現)
また、過剰発現がキシロース利用を向上させるか否かを決定するために、D−キシロース利用経路を含んでいるS. cerevisiaea株における活性な異種のD−キシロース特異的なトランスポータの過剰発現について調べた。好気性条件において振とうフラスコを用いてキシロース利用を試験した。キシローストランスポータのNCU00821、NCU04963、XUT1、STL12/XUT6、およびHxt7を発現するプラスミドを、HZE63株(CEN.PK2 ura3::キシロース利用経路)に導入した。この株は、染色体上のURA3部位に組み込まれたキシロース利用経路を有していた。それは、N. crassaに由来するキシロースリダクターゼ(XR)およびキシロースデヒドロゲナーゼ(XDH)ならびにP. stipitisに由来する(XKS)を含んでいる、これまでの研究から得られたプラスミドを用いて構築された。このプラスミドは、ApaIを用いて消化され、CEN.PK2を形質転換させて、HZE63株を生成した。
また、過剰発現がキシロース利用を向上させるか否かを決定するために、D−キシロース利用経路を含んでいるS. cerevisiaea株における活性な異種のD−キシロース特異的なトランスポータの過剰発現について調べた。好気性条件において振とうフラスコを用いてキシロース利用を試験した。キシローストランスポータのNCU00821、NCU04963、XUT1、STL12/XUT6、およびHxt7を発現するプラスミドを、HZE63株(CEN.PK2 ura3::キシロース利用経路)に導入した。この株は、染色体上のURA3部位に組み込まれたキシロース利用経路を有していた。それは、N. crassaに由来するキシロースリダクターゼ(XR)およびキシロースデヒドロゲナーゼ(XDH)ならびにP. stipitisに由来する(XKS)を含んでいる、これまでの研究から得られたプラスミドを用いて構築された。このプラスミドは、ApaIを用いて消化され、CEN.PK2を形質転換させて、HZE63株を生成した。
キシローストランスポータをコードしているプラスミドを用いて形質転換されたHZE63株を、2%のグルコースを補ったSC−Trpプレート上に播くことによって選択した。形質転換株を、2%のグルコースを有しているSC−Trp−Uraにおいて前培養し、それから0.5%または5%のキシロースを補ったSC−Trp−Uraに、最初のOD600が1.0になるまで播種した。
形質転換体の酵母のプラスミドを用いて、E. coliのpDH5α細胞を形質転換した。それから、プラスミドを単離し、確認用のPCRによって調べ、塩基配列決定に供して正しいコンストラクトであることを確認した。プラスミドマップは図39に見られる。
残念ながら、ペントース特異的なトランスポータの過剰発現の利点は、発現方法、培養条件、D−キシロース利用経路の選択の変更にもかかわらず、観察され得なかった。考えられる可能性がいくつかある。第1に、膜タンパク質(例えば糖トランスポータ)の過剰発現は、細胞膜の完全性に影響し、結果的に細胞の成長を阻害し得る(Wagner et al., 2006)。トランスポータ過剰発現株は、D−グルコースが炭素源として使用される場合でさえ、より遅い成長速度を示すことが観察された。グルコース含有のSC−ura培地において成長させた、トランスポータを有している株の2日間にわたる培養の最終的なODはわずかに4であり、ネガティブコントロールのODは約6であった。第2に、ヘキソーストランスポータを介した野生型のS. cerevisiaeのD−キシロース取込み活性は、ヘキソーストランスポータのノックアウト株において過剰発現されているD−キシローストランスポータのD−キシロース取込み活性よりはるかに高い。新たに見出されたD−キシロース特異的なトランスポータの低い糖の取込み活性は、糖の取込み能の向上を観察することを困難にさせ得る。第3に、新たなD−キシロース特異的なトランスポータの導入がS. cerevisiae細胞におけるD−キシロースの取込みを向上させ得るにしても、D−キシロース利用の利点は、D−キシロース利用経路が糖の取込みを限定的な方法にするために十分に有効である場合にのみ観察され得る。糖トランスポータの過剰発現の作用は株の背景および培養条件に依存することが示されている(Runquist et al., 2010)。後述の実施例12〜15は酵母におけるキシロース降りようの最適化を説明している。
(ペントース特異的なさらなるトランスポータのクローニング)
NCU00821、STL12/XUT6およびXUT1の相同分子種をクローン化し、ペントース取込みについて試験した。グルコースまたはペントースを補った富栄養培地において異なる真菌株を培養した。総RNAを単離し、cDNAに逆転写させた。ポリメラーゼ連鎖反応(PCR)を用いて、推定のトランスポータ遺伝子をcDNAから直接に増幅させた。しかし、調節機構および発現パターンが真菌の種におけるペントーストランスポータについて未知であったので、推定のペントーストランスポータをコードしているcDNAは、培養条件の変更にもかかわらず必ずしも入手可能ではなかった。この場合に、GenBankから提供されている対応するcDNA配列にしたがってプライマーを設計し、当該プライマーを用いてゲノムDNAを鋳型としてエキソンを増幅させた。それから、重複伸長PCRを用いてエキソンを全長遺伝子として組み合わせた。DNAアセンブラ法を用いてHXT7プロモータおよびHXT7ターミネータを含んでいるpRS424シャトルベクターに、生じたPCR産物をクローン化した。形質転換体から単離した酵母のプラスミドを用いて、E. coliDHαを形質転換し、もとのトランスポータ遺伝子を増幅するために使用されたプライマーを用いて確認用のPCRによって、単離したE. coliのプラスミドをまず検査した。
NCU00821、STL12/XUT6およびXUT1の相同分子種をクローン化し、ペントース取込みについて試験した。グルコースまたはペントースを補った富栄養培地において異なる真菌株を培養した。総RNAを単離し、cDNAに逆転写させた。ポリメラーゼ連鎖反応(PCR)を用いて、推定のトランスポータ遺伝子をcDNAから直接に増幅させた。しかし、調節機構および発現パターンが真菌の種におけるペントーストランスポータについて未知であったので、推定のペントーストランスポータをコードしているcDNAは、培養条件の変更にもかかわらず必ずしも入手可能ではなかった。この場合に、GenBankから提供されている対応するcDNA配列にしたがってプライマーを設計し、当該プライマーを用いてゲノムDNAを鋳型としてエキソンを増幅させた。それから、重複伸長PCRを用いてエキソンを全長遺伝子として組み合わせた。DNAアセンブラ法を用いてHXT7プロモータおよびHXT7ターミネータを含んでいるpRS424シャトルベクターに、生じたPCR産物をクローン化した。形質転換体から単離した酵母のプラスミドを用いて、E. coliDHαを形質転換し、もとのトランスポータ遺伝子を増幅するために使用されたプライマーを用いて確認用のPCRによって、単離したE. coliのプラスミドをまず検査した。
酵母の相同性組換えを介したDNAアセンブラ法を用いてクローニング操作のほとんを実施した。推定のペントーストランスポータのクローニングにrPS424−HXT7−GFPプラスミドを使用した。このプラスミドにおいて、HXT7ターミネータおよび両方の末端におけるEcoRI部位を用いてフランキングされたGFP遺伝子を、ClaIおよびBamHIによって直鎖化されたpRS424シャトルベクター(New England Biolabs)に組み込んだ。HXT7プロモータおよびHXT7ターミネータと同一の配列を有しているDNA断片を用いてフランキングされた、推定のペントーストランスポータのPCR産物を、EcoRI消化したpRS424−HXT7−GFPとともに、標準的な酢酸リチウム法を用いてCEN.PK2−1Cにコトランスフェクションした。2%のD−グルコースを補ったSC−Trpプレート上に、生じた形質転換混合物を播いた。それから、形質転換体をペントース輸送活性について試験した。
結果を図40および表16に示す。8つの推定のペントース特異的なトランスポータ(XP_960000(NC52)、CAG88709(DH48)、XP_457508(DH61)、XP_681669(32−10)、XP_001487429(29−6)、XP_001727326(29−9)、XP_657854(32−8)、XP_720384(29−4))のうち、NC52のみがグルコースプレートに基づく成長が可能であり、他の7つのトランスポータはペントース特異的であり得るか、または不活性であり得ることを示唆している。HPLCに基づくペントース取込みアッセイを用いて、4つのキシロース特異的なトランスポータ(XP_457508(DH61)、XP_681669(32−10)、XP_001727326(29−9)、XP_720384(29−4))が見出された。さらに、1つのアラビノース特異的なトランスポータ(XP_657854(32−8))が同定された(図40;上部)。また、さらなる5つの推定のペントース特異的なトランスポータ(XP_002488227、AB070824.1、XP_001389300、XP_002488227、EEQ43601.1)が試験され、グルコースプレートに基づいて成長可能なものはなかった。さらなるペントース取込みアッセイによって、XP_002488227およびAB070824.1はキシロース特異的なトランスポータであることが示された(図40;下部)。これらの結果の要約は表16Dに示されている。
データベースにおける配列と不一致な配列を有している相同分子種(例えば、変異を有している分子種)を、ふたたびクローン化し、配列決定し、酵母株において発現させ、糖の取込み機能について試験した。また同様に、配列決定の結果のない相同分子種をトランスポータ機能について試験した。
ペントーストランスポータの相同分子種の整列化配列を分析して、トランスポータ機能における潜在的な役割を有し得る保存されている残基を同定した。キシローストランスポータ(NCU00821、STL12/XUT6、XP_002488227.1およびXP_002382573.1)およびアラビノーストランスポータ(XUT1およびEEQ43601.1)のサンプルの整列化は、図41(a、b)にそれぞれ示されている。キシローストランスポータおよびアラビノーストランスポータの配列の全体的な比較(図41c)によって、2種類のペントーストランスポータに保存されている残基があり、ペントース取込みにおける機能的な役割を一般的に示してることがわかる。
実施例12〜15は酵母におけるキシロース利用経路の最適化に関する。
〔実施例12:ペントースを利用するS. cerevisiae株の改変〕
イソ酵素の概念を活用することによって効率的なキシロース代謝経路を再構成した。イソ酵素は、異なる動態特性または制御特性を有している同じ化学反応を触媒し、細胞基質の環境の動的な変化に応じて代謝の流れの精密に調整された制御をもたらすと知られている。しかし、イソ酵素を採用して、所望の流れを促進させる代謝改変手法はこれまで存在しなかった。本研究は、野生型および変異体のキシロースリダクターゼ(XR)の両方の同時発現が、キシロースの蓄積を低減させ得、全体のキシロース発酵速度を向上させ得ることを証明している。
イソ酵素の概念を活用することによって効率的なキシロース代謝経路を再構成した。イソ酵素は、異なる動態特性または制御特性を有している同じ化学反応を触媒し、細胞基質の環境の動的な変化に応じて代謝の流れの精密に調整された制御をもたらすと知られている。しかし、イソ酵素を採用して、所望の流れを促進させる代謝改変手法はこれまで存在しなかった。本研究は、野生型および変異体のキシロースリダクターゼ(XR)の両方の同時発現が、キシロースの蓄積を低減させ得、全体のキシロース発酵速度を向上させ得ることを証明している。
生体の系におけるイソ酵素の分布によって示唆されているように、野生型のXRおよび変異体のRX(R276H)を、キシリトールデヒドロゲナーゼ(XDH)およびキシルロキナーゼ(XK)とともに、S. cerevisiaeにおいて共発現させて、S. cerevisiaeにおいて機能的なキシロース代謝経路を構築した。XR変異体は、NADHよりはるかに低い選択性をNADPHに対して示し、野生型のXRは、NADHを超えてNADPHに対する116倍高い選択性を有していると報告されている(Watanabe et al., 2007)。
P. stipitisに由来するキシロース代謝遺伝子(野生型のXYK1、2および3ならびに変異体XYL1)をPCR増幅し、構造的なプロモータの制御下において、発現カセットを構築した。これらの組込みカセットをD452−2株のゲノムに組み込んだ。
キシロース代謝経路を構築するための発現カセットによる形質転換を、酵母EZ-Transformationキット(BIO 101, Vista, CA)を用いて実施した。アミノ酸栄養要求性マーカーを用いて形質転換体を選択するために、6.7g/リットルの酵母窒素原礎培地、20g/リットルのグルコース、20g/リットルの寒天、およびCSM−Leu−Trp−Ura(BIO 101)を含んでおり、適切なヌクレオチドおよびアミノ酸を補った酵母の合成完全(YSC)培地を使用した。20g/リットルのグルコースを有しているYP培地(10g/リットルの酵母抽出物、20g/リットルのBactoペプトン)、30℃において、酵母株を定法にしたがって培養した。
また、最適化されたキシロース利用経路の酵素を含んでいる同一のコンストラクトをいくつかの異なる酵母株において発現させることによって、キシロース代謝の効率に対するS. cerevisiae株のバックグラウンドの影響を試験した。使用した3つの研究室株は、D452−2(MATα、leu2、his3、ura3、can1)、L2612(MATa、leu2−3、leu2−112、ura3−52、trp1−298、can1、cyn1、gal+)およびCEN.PKであった。キシリトール、酢酸塩、およびエタノールの産生をキシロースおよびOD600とともに観察した。その結果は、D452−2株は3つの試験した株のなかで最も良好であった(図42〜44)。本試験に使用した株およびプラスミドを表17に記載している。
キシロースを発酵させる改変されたS. cerevisiae株は、キシロースを消費し、無視し得る量のキシリトールの蓄積をともなってエタノールを産生した。40および80g/Lのキシロースを単独の使用した場合、DA24株は、振とうフラスコおよびバイオリアクターの発酵実験の両方において、一貫した収量を有してエタノール(YEthanol/Xylose=0.31〜0.32g/g)を産生した(図45)。しかし、DA24株は、キシロースを発酵させる天然に存在する酵母P. stipitisより緩やかにキシロースを消費した。進化的な改変手法(Sauer 2001)を用いて、DA24のキシロース発酵能をさらに向上させた。キシロースを含有している培地においてDA24を繰返して継代培養した後に単離された1つの株(DA24−16)は、種々の培養条件下において親株と比べてはるかに早い発酵速度を示した(表18)。
興味深いことに、DA24−2株は、最も速やかにキシロースを発酵させる酵母であると知られているP. stipitisと同程度の速度においてキシロースを消費した。しかし、DA24によるエタノールの収率はP. stipitisよりわずかに少なかった(図46)。
ゲノムライブラリを用いて形質転換された、キシロース利用酵素を発現しているS. cerevisiae株L2612(YSX3株)を用いてスクリーニングの準備をした。形質転換の後に、キシロースの利用について効率的な株を濃縮するための酸素制限条件下の40g/Lのキシロースに継代培養を移行させた。酸素制限条件の50mLのYPX培地において発酵を実施し、OD600=10に達したときに、十分に成長した細胞培養物の0.1%(50μL)を次の継代培養に移した。10回の継代培養の後に、細胞をYPX(40g/L)寒天培地に対して段階希釈物を用いて広げた。5mLのYPX培地を用いた発酵実験を全体を通して、低いキシリトール形成および高いエタノール形成についてコロニーをスクリーニングした。DNAの配列決定によって、最も効率的な2つの株は、多コピーの組み込まれたXYL2を含んでいたことが明らかになり、それから、当該XYL2を相同性組換えによって他コピーのプラスミドにクローン化され、YSX3細胞を形質転換した。
XYL2遺伝子を、組込みベクターにおける、異なる強度のプロモータ(例えばTDHpまたはPGKp)の制御下に配置し、YSX3細胞を形質転換した(図47)。形質転換された酵母細胞におけるキシロースおよびエタノールの形成に対するこれらのプラスミドの作用を観察するために、試験を実施した。結果は、より高レベルのXYL2(PGKp下にある)を発現しているYSX3細胞は、エタノール産生においてより効率的であり、さらにより少ない量のキシリトールを産生することを示していた(図48)。さらなるXYL3がこれらの細胞において発現されている場合、産生されるキシリトールの量は、生じた株SRu−23においてさらに低下した(図49)。したがって、改変されたS. cerevisiae株におけるXYL2の発現レベルはキシロース発酵を実施するための重要な要素であり、当該株は、発現が強力なプロモータに基づく場合により少ないキシリトール蓄積を高いエタノールの収率を有していると考えられる。XYL2およびXYL3の同時の共発現によって、キシリトールの蓄積量はさらに低下する。しかし、XYL2およびXYL3を過剰発現している株においてXYL1をさらに過剰発現させた場合、相当な量のキシリトールが蓄積し、結果として低下したキシロース発酵をもたらす。したがって、効率的なキシロース発酵にとって最適なレベルのXYL1があったと考えられる。
また、XYL2およびXYL3を過剰発現しているS. cerevisiaeにおける外因性のGRE3の過剰発現がキシロース発酵を促進し得るか否かを試験するために、実験を行った。pRS403−GRE3の構築のために、GRE3遺伝子をS. cerevisiaeのD452−2から増幅させ、TDH3プロモータおよびCYCターミネータを有しているpR403ベクターに挿入した。pR403−GRE3の直鎖化の後に、D452−2のゲノムに組み込んだ。キシロース利用遺伝子を酵母株D452−2に導入し(図52)、キシロース発酵パラメータを観察した。結果は、GRE3の過剰発現は、特に、高いODの播種物として80g/Lのキシロースに基づいて細胞を成長させる場合に、エタノール産生およびキシロース蓄積におけるXYL1と同程度の効率であったことを示していた。
〔実施例13:LADおよびXDHの改変〕
NADPH依存性のXRとNAD+依存性のXDHおよびLADとの間における補助因子の不均衡によって引き起こされると考えられているL−アラビニトールおよびキシリトールの蓄積は、ペントース利用酵素を発現している改変されたS. cerevisiaeにおけるキシロース発酵の間における主要な障害と見做されている。XRおよびXDHの間における不均衡は、逆転した補助因子の選択性を有している改変された酵素によって補正されており(Watanabe et al., 2007;Matsushika et al., 2008;Bengtsson et al., 2009)、この手法は、修飾した酵素が低下した特異的活性を有しているため、低下した流れを生じた。P. stipitisのXR変異体は、NADHよりはるかに低いNADPHに対する選択性を示し、野生型のpsXRがNADPHに対して2倍の選択性を示したと報告されている(Watanabe et al., 2007)。
NADPH依存性のXRとNAD+依存性のXDHおよびLADとの間における補助因子の不均衡によって引き起こされると考えられているL−アラビニトールおよびキシリトールの蓄積は、ペントース利用酵素を発現している改変されたS. cerevisiaeにおけるキシロース発酵の間における主要な障害と見做されている。XRおよびXDHの間における不均衡は、逆転した補助因子の選択性を有している改変された酵素によって補正されており(Watanabe et al., 2007;Matsushika et al., 2008;Bengtsson et al., 2009)、この手法は、修飾した酵素が低下した特異的活性を有しているため、低下した流れを生じた。P. stipitisのXR変異体は、NADHよりはるかに低いNADPHに対する選択性を示し、野生型のpsXRがNADPHに対して2倍の選択性を示したと報告されている(Watanabe et al., 2007)。
本研究において、類似の試験がN. crassaに由来するL−アラビニトール 4−デヒドロゲナーゼ(LAD)およびXDHに対してなされて、補助因子の特異性を変更させ、したがって、改変されたS. cerevisiaeにおけるキシロース発酵を向上させた。
(推定のLADをコードしている遺伝子の同定)
LADをコードしている推定遺伝子の同定方法、ならびにLADコードしている遺伝子およびLADコードしている推定遺伝子のクローニング方法が説明されている。
LADをコードしている推定遺伝子の同定方法、ならびにLADコードしている遺伝子およびLADコードしている推定遺伝子のクローニング方法が説明されている。
推定のLADをコードしている遺伝子の同定
ncLAD(EAA36547.1)をプローブとして用いたタンパク質BLAST検索から、2つの推定遺伝子を、それぞれP. chrysogenum(XP_002569286.1)およびP. guilliermondii(EDK37120.2)において同定した。ncLADを有しているこれらの2つのタンパク質のアミノ酸配列の同一性は、それぞれ71%および46%であった。
ncLAD(EAA36547.1)をプローブとして用いたタンパク質BLAST検索から、2つの推定遺伝子を、それぞれP. chrysogenum(XP_002569286.1)およびP. guilliermondii(EDK37120.2)において同定した。ncLADを有しているこれらの2つのタンパク質のアミノ酸配列の同一性は、それぞれ71%および46%であった。
LADをコードしている遺伝子および推定のLADをコードしている遺伝子のクローニング
A. niger(NRRL 326)、P. guilliermondii(NRRL Y2075)およびP. chrysogenum(NRRL 807)を米国農務省の農業安定保全局(Peoria, IL)から入手した。T. longibrachiatum(T. reesei、YSM 768)を独国のResource Centre for Biological Material(DSMZ)から入手した。
A. niger(NRRL 326)、P. guilliermondii(NRRL Y2075)およびP. chrysogenum(NRRL 807)を米国農務省の農業安定保全局(Peoria, IL)から入手した。T. longibrachiatum(T. reesei、YSM 768)を独国のResource Centre for Biological Material(DSMZ)から入手した。
1%の酵母抽出物、2%のペプトンおよび2%のL−アラビノースを含んでいる液体培地または寒天プレートにおいて、A. niger、T. longibrachiatum、P. chrysogenumおよびP. guilliermondiiを成長させた。総RNAまたはゲノムDNAを単離するために、液体窒素において細胞を凍結させた。A. niger、T. longibrachiatum、P. chrysogenumおよびP. guilliermondiiから単離されたmRNAに対して、逆転写PCR(RT−PCR)を実施して、cDNAを入手し、PCRを用いてLAD(推定)をコードしている遺伝子を得た。A. nigerについて、推定のLAD遺伝子は、未知の理由のためにcDNAから増幅され得なかった。したがって、重複伸長PCR(OE−PCR)を用いて、単離したゲノムDNAからこのイントロンを含んでいる遺伝子をクローン化した。これらの遺伝子をクローン化するために使用されたすべてのプライマー配列は表19に挙げられている。
PCR産物をpET−28aベクターにサブクローニングし、それぞれクローニングおよび発現のために電気穿孔法によって、そのコンストラクトを用いて2つのE. coli株のDH5αおよびBL21(DE3)を形質転換した。A. niger、T. longibrachiatumおよびP. guilliermondiiから得られた予想される遺伝子のサブクローニングのためにNdeI/BamHIの制限酵素部位を使用し、P. chrysogenumから得られた予想される遺伝子のサブクローニングのためにNdeI/EcoRIの制限酵素部位を使用した。コンストラクトは、N末端のHis6−タグつきの融合体としてLAD(推定)をコードしていた。プラスミドを、BIGDYE(商標) Terminator配列決定法を用いて配列決定し、Urbana-ChampaignにあるUniversity of Illinois内のBiotechnology Center(Urbana, IL)において、3730xL Genetic Analyzer(Applied Biosystems, Foster City, CA)を用いて分析した。
タンパク質の発現および精製
pcLAD(XP_002569286.1)、pgLAD(EDK37120.2)、anLAD(CAH69383.1)およびtlLAD(AAL08944.1)をコードしている遺伝子をpET28aにクローン化し、E. coliのBL21(DE3)において発現させた。250rpm、回転式の振とう培養機、30℃において一晩にわたって、LAD遺伝子を含んでいるE. coliのBL21(DE3)を成長させた。一晩を経過した培養物(50μL)を用いて新たな培養物(5mL)に播種し、600nmにおける吸光度(OD600)が0.6〜1.0に達するまで、250rpmにおいて振とうさせながら30℃において成長させた。それから、0.3mMのIPTGを用いて、3〜5時間にわたって30℃においてか、または20時間にわたって18℃において、培養物を誘導した。
pcLAD(XP_002569286.1)、pgLAD(EDK37120.2)、anLAD(CAH69383.1)およびtlLAD(AAL08944.1)をコードしている遺伝子をpET28aにクローン化し、E. coliのBL21(DE3)において発現させた。250rpm、回転式の振とう培養機、30℃において一晩にわたって、LAD遺伝子を含んでいるE. coliのBL21(DE3)を成長させた。一晩を経過した培養物(50μL)を用いて新たな培養物(5mL)に播種し、600nmにおける吸光度(OD600)が0.6〜1.0に達するまで、250rpmにおいて振とうさせながら30℃において成長させた。それから、0.3mMのIPTGを用いて、3〜5時間にわたって30℃においてか、または20時間にわたって18℃において、培養物を誘導した。
誘導させた細胞(1mL)を、1mg/mLのリゾチームを有している1mLのリン酸カリウム緩衝液(pH7.0)にそれらを再懸濁させること、ならびに30℃および250rpmにおいて30分にわたって振とうさせることよって溶解させた。一晩にわたって80℃に細胞を維持し、室温において融解させた。生じた細胞溶解物を13200rpmにおいて15分にわたって遠心分離し、ドデシル硫酸ナトリウム−ポリアクリルアミドゲル電気泳動(SDS−PAGE)によって、タンパク質発現について上清および沈殿物を分析した。
タンパク質精製のために、誘導させた細胞(400mL)を、1mg/mLのリゾチームを有している15mLの緩衝液A(20mMのTris、0.5MのNaCl、20%のグリセロール、pH7.6)を用いて処理し、30℃および250rpmにおいて30分にわたって振とうさせた。1回の凍結融解の後に、生じた生成物を超音波処理によってさらに溶解させ、続いて12000rpmにおいて20分にわたって遠心分離して細胞片を除去した。上清をCo2+を固定化させた金属親和性クロマトグラフィー樹脂を用いて充填されているカラムにかけて、製造者の取扱説明書にしたがってHis6タグつきのタンパク質を精製した。精製したタンパク質を、限外ろ過(Amicon Ultra, Millipore, Billerica, MA)によって脱塩し、150mMのNaClおよび15%のグリセロールを含んでいるHEPES緩衝液(pH7.0)を用いて洗浄し、−20℃に維持した。タンパク質濃度を、製造者のプロトコルにしたがってBradford法(Bradford 1976)によって決定した。
(LADタンパク質の性質決定)
LAD酵素の、定常状態の動態、分子量、四次構造、温度依存性、pH依存性、L−アラビニトールデヒドロゲナーゼ活性、および金属含有量を分析した。
LAD酵素の、定常状態の動態、分子量、四次構造、温度依存性、pH依存性、L−アラビニトールデヒドロゲナーゼ活性、および金属含有量を分析した。
L−アラビニトールデヒドロゲナーゼ活性
P. chrysogenum、P. guilliermondii、A. nigerおよびT. longibrachiatumに由来するLADを発現している宿主細胞から溶解物を調製した。50mMのリン酸カリウム緩衝液(pH7.0)における基質として200mMのL−アラビニトールおよび2mMのNAD+を用いた活性アッセイのために、10マイクロリットルの細胞溶解物を使用した。Cary 300 Bio UV-vis分光光度計(Varian, Cary, NC)を用いて340nmにおける吸光度(ε=6.22mM−1cm−1)を測定することによって、NADH産生を観察した。
P. chrysogenum、P. guilliermondii、A. nigerおよびT. longibrachiatumに由来するLADを発現している宿主細胞から溶解物を調製した。50mMのリン酸カリウム緩衝液(pH7.0)における基質として200mMのL−アラビニトールおよび2mMのNAD+を用いた活性アッセイのために、10マイクロリットルの細胞溶解物を使用した。Cary 300 Bio UV-vis分光光度計(Varian, Cary, NC)を用いて340nmにおける吸光度(ε=6.22mM−1cm−1)を測定することによって、NADH産生を観察した。
定常状態の動態
異なるLAD酵素の動態パラメータを決定した。50mMのリン酸カリウム緩衝液(pH7.0)および室温においてUV-vis分光光度計を用いて340nmにおける吸収の変化を測定することによって、初期の割合を決定した。種々の濃度の基質(L−アラビニトール)および補助因子(NAD+/NADP+)(L−アラビニトールについて5〜320mM、補助因子について0.5mM〜3.2mM)における初期の割合を測定した。ミカエリス−メンテンの反応速度論を用いて基質および補助因子についての酵素動態を分析し、ラインウィーバー−バークのプロットにデータを適合させることによって動態パラメータを決定した。飽和した補助因子の濃度(3.2mM)における初期の割合を測定することによって基質についてのパラメータを決定し、飽和した基質の濃度(320mM)において補助因子についてのパラメータを決定した。アッセイを3組において実施した。
異なるLAD酵素の動態パラメータを決定した。50mMのリン酸カリウム緩衝液(pH7.0)および室温においてUV-vis分光光度計を用いて340nmにおける吸収の変化を測定することによって、初期の割合を決定した。種々の濃度の基質(L−アラビニトール)および補助因子(NAD+/NADP+)(L−アラビニトールについて5〜320mM、補助因子について0.5mM〜3.2mM)における初期の割合を測定した。ミカエリス−メンテンの反応速度論を用いて基質および補助因子についての酵素動態を分析し、ラインウィーバー−バークのプロットにデータを適合させることによって動態パラメータを決定した。飽和した補助因子の濃度(3.2mM)における初期の割合を測定することによって基質についてのパラメータを決定し、飽和した基質の濃度(320mM)において補助因子についてのパラメータを決定した。アッセイを3組において実施した。
クローン化したLADは、L−アラビニトールに対する異なる結合親和性および触媒活性を示した(LADのなかでもKmは2倍ほど異なっており、kcatは3倍ほど異なっていた)。L−アラビニトールについて、anLAD、tlLADおよびpcLADのKm値は、それぞれ25±1、18±1および37±2mMであり、kcat値は、507±22、346±41および1085±71分−1であった(表20)。tlLAD酵素は最も低いを有しており、pcLADは、最も高いKmを有しているにもかかわらず、最も高い触媒活性(kcat)および効率(kcat/Km)を示した(表20)。補助因子のNAD+の動態について、クローン化したLADは0.2〜0.3mMの範囲におけるKm値を示し、2526〜3460mM−1分−1の範囲における触媒効率を示した(表21)。クローン化したすべてのLADは、NADP+に対して最小の活性を示した(表20、21)。初期の割合は、高いKmのために、最も高い基質濃度および補助因子濃度(320mMのL−アラビニトールおよび3.2mMのNADP+)において飽和しなかった。したがって、0.1もしくは0.2mMのNADP+および10もしくは20mMのL−アラビニトールを用いて(Km>>[S])、酵素について触媒効率のみを決定した(表20、21)。
分子量および四次構造
4つのタンパク質のサブユニットの算出された分子量は、43kDa(anLAD)、41kDa(tlLAD)、42kDa(pcLAD)および42kDa(pgLAD)であった。Shimadzu HPLCシステム(Shimadzu, Kyoto, Japan)におけるBio-Sil SEC-250 column(300×7.8mm、Bio-Rad, Hercules, CA)を用いて、タンパク質の分子量を決定した。移動相は、50mMのNa2HPO4、50mMのNaH2PO4、150mMのNaClおよび10mMのNaN3(pH6.8)からなり、流速は1.0mL/分であった。タンパク質の分子量基準と滞留時間とを比較することによって、分子量を算出した。
4つのタンパク質のサブユニットの算出された分子量は、43kDa(anLAD)、41kDa(tlLAD)、42kDa(pcLAD)および42kDa(pgLAD)であった。Shimadzu HPLCシステム(Shimadzu, Kyoto, Japan)におけるBio-Sil SEC-250 column(300×7.8mm、Bio-Rad, Hercules, CA)を用いて、タンパク質の分子量を決定した。移動相は、50mMのNa2HPO4、50mMのNaH2PO4、150mMのNaClおよび10mMのNaN3(pH6.8)からなり、流速は1.0mL/分であった。タンパク質の分子量基準と滞留時間とを比較することによって、分子量を算出した。
HPLCによって観察された分子量およびSDS−PAGE分析によって決定された単量体サブユニットの分子量に基づいて、四次構造を決定した。an−、tl−およびpcLADの分子量をそれぞれ178、194および173kDaと決定した。SDS−PAGEによって決定したサブユニットの分子量との比較によって、結果は、LADが未変性の形態において非共有結合的に連結された四量体であることを示した。
温度依存性およびpH依存性
10〜70℃の範囲の温度において酵素活性をアッセイすることによって、タンパク質の至適温度を決定した。50℃のリン酸緩衝液における種々のインキュベーション時間の後における酵素活性を測定することによって、熱不活性化を決定した。2mMのNAD+および200mMのL−アラビニトールを用いて酵素活性を測定した。一次の指数関数的な減衰関数を用いて酵素活性の半減期を決定した。UV-vis分光光度計に接続されたCary温度制御器(Varian, Cary, NC)によって温度を制御した。一般的な緩衝液(50mMのモルホリンエタンスルホン酸/50mMのTris/50mMのグリシン)(Ellis and Morrison 1982)、飽和濃度のNAD+(2mM)およびL−アラビニトール(200mM)、5.0〜11.0の間のpHにおいて活性を測定することによって、温度依存性の酵素活性を決定した。
10〜70℃の範囲の温度において酵素活性をアッセイすることによって、タンパク質の至適温度を決定した。50℃のリン酸緩衝液における種々のインキュベーション時間の後における酵素活性を測定することによって、熱不活性化を決定した。2mMのNAD+および200mMのL−アラビニトールを用いて酵素活性を測定した。一次の指数関数的な減衰関数を用いて酵素活性の半減期を決定した。UV-vis分光光度計に接続されたCary温度制御器(Varian, Cary, NC)によって温度を制御した。一般的な緩衝液(50mMのモルホリンエタンスルホン酸/50mMのTris/50mMのグリシン)(Ellis and Morrison 1982)、飽和濃度のNAD+(2mM)およびL−アラビニトール(200mM)、5.0〜11.0の間のpHにおいて活性を測定することによって、温度依存性の酵素活性を決定した。
anLADおよびpcLADの至適温度は40〜50℃の間にあり、tlLADは55〜65℃の範囲のより高い至適温度を示した(図55a)。LADの触媒活性は、50℃におけるインキュベーション時間の長さにしたがって指数的に低下し、100分後にほぼ完全に不活性化された(図55b)。tlLADは、50℃において20分間の半減期を有して最も熱的に安定であり、anLADは、50℃において20分未満の半減期を有して最も不安定であった。性質決定されたすべてのLADは、約pH9.4において最大の活性を有して、7〜11のpH範囲において活性を示した(図55c)。9〜10を外れるpH範囲において活性は有意に低下し、20%の活性がpH7.0において残っていた(図55c)。pH5.0未満において活性は検出されなかった。
金属分析
緩衝液交換および凍結乾燥によって、金属分析のための2組のサンプルを、リン酸緩衝液(PBS)において調製した。各サンプルは1mLの緩衝溶液に1〜2mgのタンパク質を含んでいた。Urbana-Champaignにあるイリノイ大学のMicroanalytical Laboratory(Urbana, IL)における誘導結合高周波プラズマ原子発光分析(OES Optima 2000 DV, Perkin Elmer, Boston, MA)によって、金属の同一性および含有量を分析した。
緩衝液交換および凍結乾燥によって、金属分析のための2組のサンプルを、リン酸緩衝液(PBS)において調製した。各サンプルは1mLの緩衝溶液に1〜2mgのタンパク質を含んでいた。Urbana-Champaignにあるイリノイ大学のMicroanalytical Laboratory(Urbana, IL)における誘導結合高周波プラズマ原子発光分析(OES Optima 2000 DV, Perkin Elmer, Boston, MA)によって、金属の同一性および含有量を分析した。
測定されたZn2+の重量%は、1:1のモル比に基づいて算出された重量%と近かった(表22)。
(変更された補助因子特異性を有しているLAD酵素の改変)
LADの補助因子特異性を変更させる方法が決定され、変異生成されたLADは、変更された補助因子特異性および他の特性について分析された。
LADの補助因子特異性を変更させる方法が決定され、変異生成されたLADは、変更された補助因子特異性および他の特性について分析された。
変更された補助因子特異性を有しているLAD酵素の生成
anLAD、tlLADおよびpcLADの補助因子特異性をNAD+からNADP+に変更させるために、部位特異的な変異生成を実施した。天然に存在するtlLADのアミノ酸番号の224、225および362のそれぞれを、セリン、アルギニンおよびスレオニンに置換して、変更された補助因子特異性を有しているtlLADを生成させた。クローン化されたanLADおよびpcLADのアミノ酸配列を、T. longibrachiatumのLAD(tlLAD)配列と整列化させ、tlLADのアミノ酸番号の224、225および362に対応するアミノ酸を変異させた。変更された補助因子特異性を有しているLADのすべてについて、補酵素結合ドメインのβ−α−βモチーフ内の2つのアミノ酸残基(amLADのD213およびI214、tlLADのD224およびI225、ならびにpcLADのD212およびI213)をそれぞれセリンおよびアルギニンに置き換え(Korkhin et al., 1998; Pauly et al., 2003; Watanabe et al., 2005)、3番目の変異を、anLADのA359、tlLADのA362、pcLADのS358に導入して、スレオニンに置換した(プライマー配列については表23を参照)。メガプライマーPCR法を使用して、鋳型として野生型のLADコンストラクトを用いて部位特異的な変異を導入した(Sarkar and Sommer 1990)。DNA配列分析によって正しい変異生成を確認した。
anLAD、tlLADおよびpcLADの補助因子特異性をNAD+からNADP+に変更させるために、部位特異的な変異生成を実施した。天然に存在するtlLADのアミノ酸番号の224、225および362のそれぞれを、セリン、アルギニンおよびスレオニンに置換して、変更された補助因子特異性を有しているtlLADを生成させた。クローン化されたanLADおよびpcLADのアミノ酸配列を、T. longibrachiatumのLAD(tlLAD)配列と整列化させ、tlLADのアミノ酸番号の224、225および362に対応するアミノ酸を変異させた。変更された補助因子特異性を有しているLADのすべてについて、補酵素結合ドメインのβ−α−βモチーフ内の2つのアミノ酸残基(amLADのD213およびI214、tlLADのD224およびI225、ならびにpcLADのD212およびI213)をそれぞれセリンおよびアルギニンに置き換え(Korkhin et al., 1998; Pauly et al., 2003; Watanabe et al., 2005)、3番目の変異を、anLADのA359、tlLADのA362、pcLADのS358に導入して、スレオニンに置換した(プライマー配列については表23を参照)。メガプライマーPCR法を使用して、鋳型として野生型のLADコンストラクトを用いて部位特異的な変異を導入した(Sarkar and Sommer 1990)。DNA配列分析によって正しい変異生成を確認した。
本実施例において“tlLAD変異体”は、変異D224S/I225R/A362Tを有しているtlLADと定義づけられ、“anLAD変異体”は、変異D213S/I214R/A362Tを有しているanLADと定義づけられ、“pcLAD変異体”は、変異D212S/I213R/S358Tを有しているpcLADと定義づけられる。tlLAD変異体は、NAD+からNADP+へ有意に変更された補助因子特異性を示した。また、それは最も高い触媒活性について証明された。NADP+を有しているL−アラビニトールに対するtlLAD変異性のKmおよびkcatはそれぞれ46±4mMおよび170±9分−1であった(表24)。飽和したNAD+とともにtlLAD変異体を含んでいるすべてのアッセイにおいて、反応速度のプラトーは、試験された濃度範囲において観察されず、したがって、触媒効率は、NAD+に対して0.8mMおよびL−アラビニトールに対して80mMと決定された(表24、25)。補助因子について、anLAD変異体およびpcLAD変異体は、NAD+を超えてNADP+に対する有意に高い選択性を示した(表25)。anLAD変異体およびpcLAD変異体のKm値は0.46±0.09および0.10±0.01mMであり、anLAD変異体およびpcLAD変異体のkcat値は55.7±6.4および90.5±9.2分−1であった。anLAD変異体およびpcLAD変異体の触媒効率は130±32および934±72mM−1分−1であり、NAD+に対するNADP+の触媒効率の割合は100および161であった。tlLAD変異体について、NAD+に対するNADP+の触媒効率の割合は2.5×104倍まで上昇した(表21、25)。pcLAD変異体はNAD+に対する活性を示さなかった。
(変更された補助因子特異性を有しているN. crassaのXDH(ncXDH)の改変)
推定のncXDHのクローニングおよび性質決定
P. stipitisのキシリトールデヒドロゲナーゼ(psXDH)酵素をクエリ配列として用いたNational Center for Biotechnology Informationのウェブサイト(ウェブページncbi.nlm.nih.gov)におけるタンパク質BLAST検索を用いて、N. crassaの推定のキシリトールデヒドロゲナーゼ(ncXDH)の配列を見出した。ClustalWの演算手順を用いて2つの酵素を完全に整列化させ、44%の同一性および60%の類似性を共有していることを見出した(図56)。Neurospora crassaの全体のゲノム配列が公開されており(Galagan et al., 2003)、当該ゲノム配列を推定のキシリトールデヒドロゲナーゼ(XDH)遺伝子のクローニング用のプライマーの設計に利用した。
推定のncXDHのクローニングおよび性質決定
P. stipitisのキシリトールデヒドロゲナーゼ(psXDH)酵素をクエリ配列として用いたNational Center for Biotechnology Informationのウェブサイト(ウェブページncbi.nlm.nih.gov)におけるタンパク質BLAST検索を用いて、N. crassaの推定のキシリトールデヒドロゲナーゼ(ncXDH)の配列を見出した。ClustalWの演算手順を用いて2つの酵素を完全に整列化させ、44%の同一性および60%の類似性を共有していることを見出した(図56)。Neurospora crassaの全体のゲノム配列が公開されており(Galagan et al., 2003)、当該ゲノム配列を推定のキシリトールデヒドロゲナーゼ(XDH)遺伝子のクローニング用のプライマーの設計に利用した。
D−キシロースによって誘導されたN. crassaの10333から単離された総RNAに対して実施されたRT−PCRは、予想されたサイズの遺伝子の生成物(〜1.1kb)を示した。NdeIおよびSacIの制限酵素部位を用いて、RT−PCRの産物をpET−28aベクターにクローン化し、E. coliのBL21(DE3)を形質転換した。このコンストラクト(pET−28a ncXDH)は、トロンビン切断部位とともにN末端のHis6−タグつきの融合物としてncXDHを発現した。これらの細胞のIPTG誘導した培養物の溶解物を調製し、SDS−PAGEによって分析し、XDH活性についてアッセイした。それから、製造者のプロトコルにしたがってTalon(登録商標) Co2+ Superflow樹脂(Clontech, Mountain View, CA)を用いた固定化された金属イオンの親和性クロマトグラフィー(IMAC)によってXDHを精製した。15%のグリセロールを加えた50mMの4−(2−ヒドロキシエチル)ピペラジン−1−エタンスルホン酸(HEPES)緩衝液の数回にわたる洗浄をともなう限外ろ過によって精製されたタンパク質を、脱塩し、−80℃において凍結させて保存した。タンパク質濃度をBradford法(Bradford 1976)によって決定した。
ncXDHは忠実にNAD+を選択する酵素である。また、ncXDHは高い安定性(50℃における〜200分間の半減期)および発現を示した。Watanabe et al. (2005b)によるこれまでの研究はpsXDHの補助因子特異性を逆転させることを目的としていた。
変更された補助因子特異性を有しているncXDHの生成
配列の整列化を介して、ncXDHのD204、I205およびV206のそれぞれをアラニン、アルギニンおよびセリンへの部位特異的な変異生成のために標的化して、ncXDH−ARSを生成した。表26は、ncXDH−ARSが、完全に逆転された補助因子特異性を有しており、NADP+を選択することを示している。
配列の整列化を介して、ncXDHのD204、I205およびV206のそれぞれをアラニン、アルギニンおよびセリンへの部位特異的な変異生成のために標的化して、ncXDH−ARSを生成した。表26は、ncXDH−ARSが、完全に逆転された補助因子特異性を有しており、NADP+を選択することを示している。
ncXDH変異体の動態
変異体ncXDHは、補助因子特異性の顕著な反転を有している。NADP+に対する変異体ncXDHのKmは、NAD+に対する野生型のncXDHのKmより約2.5倍だけ高く、kcat値も同様であった(表27)。
変異体ncXDHは、補助因子特異性の顕著な反転を有している。NADP+に対する変異体ncXDHのKmは、NAD+に対する野生型のncXDHのKmより約2.5倍だけ高く、kcat値も同様であった(表27)。
図57に示されているように、XDH活性は、pH4.0に至るまで活性を有しており、より酸性の条件に対するより高い耐性を示し、LAD活性はインビトロ活性アッセイにおいてpH5.0において破壊される。
〔実施例14:Bacteroides stercoris由来のキシロースイソメラーゼの、S. cerevisiaeにおける発現〕
細菌のキシロースイソメラーゼ(XI)はキシロースのキシルロースへの変換に関与する。近年、嫌気性真菌(Piromyces sp.およびOrpinomyces sp.)の2つの種、および嫌気性細菌(Clostridium phytofermentans)から活性なXIを発現させることに成功した3つの事例が報告されている。Piromyces sp.のE2に由来する真菌のXYLA遺伝子はS. cerevisiaeにおいて機能的に発現されており、最大で1.1U/1mgのタンパク質のXI活性が30℃において得られた(Kuyper et al., 2003)。また、Piromyces sp.に由来する遺伝子と94%の同一性を有している、Orpinomycesに由来する他の真菌のXYLA遺伝子は、S. cerevisiaeにおいて機能的に発現されていた(Madhavan et al., 2009)。近年において、Clostridium phytofermentansに由来する第1の原核生物のxylA遺伝子はS. cerevisiaeにおいて機能的に発現された(Brat et al., 2009)。
細菌のキシロースイソメラーゼ(XI)はキシロースのキシルロースへの変換に関与する。近年、嫌気性真菌(Piromyces sp.およびOrpinomyces sp.)の2つの種、および嫌気性細菌(Clostridium phytofermentans)から活性なXIを発現させることに成功した3つの事例が報告されている。Piromyces sp.のE2に由来する真菌のXYLA遺伝子はS. cerevisiaeにおいて機能的に発現されており、最大で1.1U/1mgのタンパク質のXI活性が30℃において得られた(Kuyper et al., 2003)。また、Piromyces sp.に由来する遺伝子と94%の同一性を有している、Orpinomycesに由来する他の真菌のXYLA遺伝子は、S. cerevisiaeにおいて機能的に発現されていた(Madhavan et al., 2009)。近年において、Clostridium phytofermentansに由来する第1の原核生物のxylA遺伝子はS. cerevisiaeにおいて機能的に発現された(Brat et al., 2009)。
嫌気性細菌Bacteroides stercorisに由来するイソメラーゼ遺伝子xylA(BtXI)は、Piromyces sp.に由来するイソメラーゼ遺伝子と高い同一性を共有している(82%)。BtXIをpRS424TEFベクターにクローン化し、S. cerevisiaeL2612株を形質転換させた。また、pRS403TEFベクターを用いることによって、S. cerevisiaeD452−2株に上記遺伝子を組み込んだ。BtXIを発現している両方の株におけるエタノール産生(L2612において5g/LおよびD452−2において7.8g/L)を観察した(図58〜59)。しかし、産生の速度は、XYL遺伝子を発現している改変された株の速度と比べて、相対的に低かった。
低いエタノール産生は、蓄積した任意のキシリトール(内因性の酵母のアルドースリダクターゼによってキシロースから形成された)の抑制性の作用のためであり得る。キシリトール蓄積を減少させるために、BTXIを発現している酵母株においてXDHおよびXKを発現させた(DBtXI)。生じた株はわずかに向上したエタノール産生および低下したキシリトール産生を示した(図60)。DBtXIにおけるこれらの2つのXYL遺伝子の共発現は好気性条件下においてさえエタノール産生を起こした。
〔実施例15:ペントースリン酸化経路(PPP)における酵素の過剰発現〕
P. stipitisに由来するPPP酵素グルコース−6−リン酸デヒドロゲナーゼ(ZWF1)、6−ホスホグルコン酸デヒドロゲナーゼ(GDN1)、トランスアルドラーゼ(TAL1)、およびトランスケトラーゼ(TKT1)を、強力なプロモータ(PGPD)の制御下において組込みベクター(pRS406)にクローン化した。そのプラスミドを酵素StuIによって直鎖化し、S. cerevisiaeの染色体に組み込んだ。
P. stipitisに由来するPPP酵素グルコース−6−リン酸デヒドロゲナーゼ(ZWF1)、6−ホスホグルコン酸デヒドロゲナーゼ(GDN1)、トランスアルドラーゼ(TAL1)、およびトランスケトラーゼ(TKT1)を、強力なプロモータ(PGPD)の制御下において組込みベクター(pRS406)にクローン化した。そのプラスミドを酵素StuIによって直鎖化し、S. cerevisiaeの染色体に組み込んだ。
しかし、PPP酵素を過剰発現させる有益な作用を得るために、XYL3(XK)を過剰発現させる必要があった(図61)。また、XYL3およびPPP酵素はYPキシルロース培地におけるエタノール産生を向上させた。
〔実施例16:アルドース−1−エピメラーゼの発現〕
β−グルコシダーゼによるセロビオースの加水分解はβ−D−グルコースを放出する。しかし、酵母のヘキソキナーゼはα−D−グルコースを選択し(または排他的に利用し)、α−D−グルコースに対するβ−D−グルコースの変旋光の割合は、代謝速度を実質的に低下させる。変換を促進させる1つの方法は、推定のアルドース−1−エピメラーゼNCU09705を過剰発現させることであった。この仮説は、NCU09705の相同物:E. coliにおけるgalM;S. cerevisiaeにおけるGAL10、YHR210CおよびYNR071C;ならびにP. stipitisにおけるGAL10を過剰発現させることによって試験された。それから、株は、セロビオース消費およびエタノール産生について試験された(図62)。結果は、S. cerevisiaeにおける相同物の過剰発現がセロビオース消費およびエタノール産生のわずかな向上を起こさせることを示した。
β−グルコシダーゼによるセロビオースの加水分解はβ−D−グルコースを放出する。しかし、酵母のヘキソキナーゼはα−D−グルコースを選択し(または排他的に利用し)、α−D−グルコースに対するβ−D−グルコースの変旋光の割合は、代謝速度を実質的に低下させる。変換を促進させる1つの方法は、推定のアルドース−1−エピメラーゼNCU09705を過剰発現させることであった。この仮説は、NCU09705の相同物:E. coliにおけるgalM;S. cerevisiaeにおけるGAL10、YHR210CおよびYNR071C;ならびにP. stipitisにおけるGAL10を過剰発現させることによって試験された。それから、株は、セロビオース消費およびエタノール産生について試験された(図62)。結果は、S. cerevisiaeにおける相同物の過剰発現がセロビオース消費およびエタノール産生のわずかな向上を起こさせることを示した。
〔実施例17:キシロースおよびセロビオースの共発酵〕
本実施例において、新たな方法を用いて、グルコースの二量体セロビオースがキシロースとともに共発酵されるときのグルコース抑制を解消させた。セロビオースは、セルロースの酵素的な加水分解からの中間生成物であり、セルラーゼ(エキソセルラーゼ、エンドセルラーゼおよびβ−グルコシダーゼが挙げられる)のカクテルにおけるβ−グルコシダーゼによってグルコースにさらに変換される。ペントース糖は、ヘミセルロースの希酸加水分解の生成物である。野生型のS. cerevisiaeは、セロビオーストランスポータおよびセロビオースをグルコースに加水分解可能なβ−グルコシダーゼの両方を欠いているため、セロビオースを消化できない。実施例9に記載の新たに見出されたセロデキストリントランスポータ遺伝子、およびN. crassa由来のβ−グルコシダーゼ遺伝子がS. cerevisiaeにおいて共発現されると、キシロースおよびセロビオースの混合物は、炭素源として利用された(図63)。類似の手法は、S. cerevisiaeによるセロビオース発酵を可能にさせるために、β−グルコシダーゼの分泌または細胞表面における提示のいずれかを採用している(van Rooyen et al., 2005;Skory et al., 1996;Kotaka et al., 2008;Katahira et al., 2006)。これらの場合、セロビオースは、S. cerevisiaeの内因性のヘキソース輸送系よって輸送される前に、細胞外においてグルコースに加水分解された。対照的に、この方法において、セロビオースは輸送に続いて細胞内において加水分解された。
本実施例において、新たな方法を用いて、グルコースの二量体セロビオースがキシロースとともに共発酵されるときのグルコース抑制を解消させた。セロビオースは、セルロースの酵素的な加水分解からの中間生成物であり、セルラーゼ(エキソセルラーゼ、エンドセルラーゼおよびβ−グルコシダーゼが挙げられる)のカクテルにおけるβ−グルコシダーゼによってグルコースにさらに変換される。ペントース糖は、ヘミセルロースの希酸加水分解の生成物である。野生型のS. cerevisiaeは、セロビオーストランスポータおよびセロビオースをグルコースに加水分解可能なβ−グルコシダーゼの両方を欠いているため、セロビオースを消化できない。実施例9に記載の新たに見出されたセロデキストリントランスポータ遺伝子、およびN. crassa由来のβ−グルコシダーゼ遺伝子がS. cerevisiaeにおいて共発現されると、キシロースおよびセロビオースの混合物は、炭素源として利用された(図63)。類似の手法は、S. cerevisiaeによるセロビオース発酵を可能にさせるために、β−グルコシダーゼの分泌または細胞表面における提示のいずれかを採用している(van Rooyen et al., 2005;Skory et al., 1996;Kotaka et al., 2008;Katahira et al., 2006)。これらの場合、セロビオースは、S. cerevisiaeの内因性のヘキソース輸送系よって輸送される前に、細胞外においてグルコースに加水分解された。対照的に、この方法において、セロビオースは輸送に続いて細胞内において加水分解された。
S. cerevisiaeにおける混合糖の発酵のための従来の方法において、リグノセルロースに由来するグルコースおよびペントース糖の混合物が使用されている。しかし、この新しい方法においてセロビオースおよびペントース糖が使用された。セロビオースは、異種のセロデキストリントランスポータによって酵母細胞の内部に輸送され、ペントースは、内因性のヘキソーストランスポータによって酵母細胞の内部に輸送され、したがって、グルコース抑制を部分的に担っている現象である同じトランスポータにとってのグルコースおよびペントースの間における直接の競合を排除している。いったん酵母細胞の内部に入ると、セロビオースは、β−グルコシダーゼによってグルコースに変換され、酵母細胞によって直ちに消費され、細胞内の低いグルコース濃度を生じることによって、グルコース抑制をさらに軽減する。
キシロースを利用する改変された酵母株L2612を、セロデキストリントランスポータおよびβ−グルコシダーゼ遺伝子を共発現する宿主として使用した。この株において、Pichia stipitisに由来するキシロースリダクターゼ、キシリトールデヒドロゲナーゼおよびキシルロキナーゼからなるD−キシロース利用経路が染色体に組み込まれている。Neurospora crassaに由来するセロデキストリントランスポータ(NCU00821、NCU08114およびNCU00809が挙げられる)、ならびに2つのβ−グルコシダーゼ遺伝子、Neurospora crassaに由来するβ−グルコシダーゼ遺伝子およびAspergillus aculeatusに由来するβ−グルコシダーゼ遺伝子を評価した。
S. cerevisiaeのL2612(MATα、leu2−3、leu2−112、ura3−52、trp1−298、can1、cyn1、gal+)を、合成のドロップアウト培地(アミノ酸および硫酸アンモニウムなしの0.17%のDifco酵母窒素原礎培地、0.5%の硫酸アンモニウム、0.05%のアミノ酸のドロップアウト混合物)において培養して、プラスミドを維持させた。2%の糖を有しているYPA培地(1%の酵母抽出物、2%のペプトン、0.01%のヘミ硫酸アデニン)を用いて酵母株を成長させた。
Pichia stipitisに由来するD−キシロースリダクターゼ、キシリトールデヒドロゲナーゼおよびキシルロキナーゼからなるD−キシロース利用経路を組み込むために、対応する遺伝子を、PCRによって増幅させ、DNAアセンブラ法(Shao et al., 2009)を用いてpRS416プラスミドにクローン化した。BamHIおよびHindIIIを使用して、D−キシロース利用経路をコードしているDNA断片を取り出し、それから2つの同じ制限酵素によって消化したpRS406プラスミドに連結した。それから、生じたプラスミドをApaIによって直鎖化し、L2612の染色体上のURA3遺伝子座に組み込んだ。
pRS425プラスミド(New England Biolabs, Ipswich, MA)を使用して、セロデキストリントランスポータ遺伝子およびβ−グルコシダーゼ遺伝子を共発現させた。図64に示されているように、pRS425プラスミドをBamHIおよびApaIによって消化した。セロデキストリントランスポータのN末端およびC末端にそれぞれPYK1プロモータおよびADHターミネータを加え、β−グルコシダーゼのN末端およびC末端にそれぞれTEF1プロモータおよびPGL1ターミネータを加えた。DNAアセンブラ法(Shao et al., 2009)を用いてこれらの遺伝子断片を、直鎖化したpRS425シャトルベクターに組み合わせた。3つのセロデキストリントランスポータ遺伝子、Neurospora crassaに由来するNCU00801(XM_958708)、NCU08114(XM_958780)およびNCU00809(XM_959259)、ならびに2つのβ−グルコシダーゼ遺伝子、Neurospora crassaに由来するNCU00130(yy)およびAspergillus aculeatusに由来するBGL1(D64088)を使用した。
それから、酵母のプラスミドをE. coliDH5αに移し、組換えDNA操作のために使用した。00mg/Lのアンピシリンを含んでいるLuriaブロスプレートに形質転換体を播いた。それから、E. coli形質転換体の単一のコロニーをLuriaブロス培地(Fisher Scientific, Pittsburgh, PA)に播種し、37℃および250rpmにおいて成長させた。QIAprep Spin Miniprep Kit(QIAGEN)を用いてプラスミドをE. coliから単離した。これらのプラスミドを用いてL2612株を形質転換して、以下の株:SL01(Neurospora crassaに由来するセロデキストリントランスポータ遺伝子NCU00801およびβ−グルコシダーゼ遺伝子NCU00130を有しているプラスミドを含んでいる)、SL02(Neurospora crassaに由来するセロデキストリントランスポータ遺伝子NCU00809およびβ−グルコシダーゼ遺伝子NCU00130を有しているプラスミドを含んでいる)、SL03(Neurospora crassaに由来するセロデキストリントランスポータ遺伝子NCU08117およびβ−グルコシダーゼ遺伝子NCU00130を有しているプラスミドを含んでいる)、SL04(セロデキストリントランスポータ遺伝子NCU00801およびAspergillus aculeatusに由来するBGL1遺伝子を有しているプラスミドを含んでいる)、SL05(セロデキストリントランスポータ遺伝子NCU00809およびAspergillus aculeatusに由来するBGL1遺伝子を有しているプラスミドを含んでいる)、およびSL06(セロデキストリントランスポータ遺伝子NCU08114およびAspergillus aculeatusに由来するBGL1遺伝子を有しているプラスミドを含んでいる)をそれぞれ生じさせた。空のpRS425プラスミドを用いてL2612株を形質転換させて、SL00株を生じさせ、ネガティブコントロールとして使用した。標準的な酢酸リチウム法(Gietz et al., 1995)を用いて酵母の形質転換を実施した。2%のD−グルコースを補ったSC−Ura−Leu培地に、生じた形質転換体の混合物を播いた。
DNAアセンブラ法を用いてプラスミドの適切な構成を確認するために、Zymoprep Yeast Plasmid Miniprep IIキット(Zymo Research, Orange, CA)を用いて、酵母細胞からプラスミドを単離し、それから、E. coliのDH5α細胞に移した。100mg/Lのアンピシリンを含んでいるLBプレートに生じた細胞を播いた。E. coliの単一のコロニーをLB液体培地に播種した。QIAprep Spin Miniprepキット(QIAGEN, Valencia, CA)を用いて、E. coliからプラスミドを単離し、確認用のPCR、またはClaIおよびHindIIIを用いた制限酵素消化によって確認した。すべての制限酵素をNew England Biolabs(Ipswich, MA)から購入した。すべての化学薬品をSigma AldrichまたはFisher Scientificから購入した。
各酵母株について、2%のグルコースを含んでいる2mLのSC−Ura−Leu培地において単一のコロニーをまず成長させ、それから、250mLの振とうフラスコに入っている50mLの同じ培地に播種して、混合糖の発酵試験のために十分な細胞を得た。成長の1日後に、細胞を遠心沈殿させた。それから、4%のセロビオースおよび5%のD−キシロース;4%のセロビオース、5%のキシロースおよび0.5%のグルコース;または4%のセロビオース、5%のキシロース、および1%のグルコースを補った、250mLのバッフルのない振とうフラスコに入っている50mLのYP培地に細胞を播種した。1以下の初期のOD600から開始して、細胞培養物を、酸素制限条件下の発酵のために30℃および100rpmにおいて成長させた。OD600の読み取りおよび細胞培養物のサンプルの取得を種々の時点において行った。HPLCを用いて糖濃度を分析し、Ethanolキット(R-biopharm, Darmstadt, Germany)を用いてエタノール産生を分析した。各データポイントについて、3組のサンプルを利用した。SL00〜SL06の株についての混合糖の発酵の結果を図65に示す。最良の株SL01をさらなる性質決定のために選択した。
1つのセロデキストリントランスポータ遺伝子および1つのβ−グルコシダーゼ遺伝子を有しているpRS425プラスミドをL2612株に導入することによって、SL01〜SL06の6つの異なる株のすべてを構築した。各プラスミドにおいて、セロデキストリントランスポータ遺伝子およびβ−グルコシダーゼ遺伝子を、酵母のプロモータおよびターミネータをとともに加え、DNA10アセンブラ法(Shao et al., 2009)によって多コピーのプラスミドpRS425に組み込んだ(図64)。空のpRS425プラスミドをL2612株に導入して、ネガティブコントロールとして使用したSL00株を生成した。40g/Lのセロビオースおよび50g/LのD−キシロースの混合物とともに振とうフラスコにおいて、すべての株を培養し、それらの糖の消費速度、細胞増殖速度、エタノール力価を決定した(図65)。すべての株のうち、Neurospora crassaに由来するβ−グルコシダーゼおよびセロデキストリントランスポータNCU00801を含んでいるSL01株は、最も高い糖の消費速度、およびエタノール生産性を示した。したがってこの株をさらなる性質決定のために選択した。
振とうフラスコおよびバイオリアクターの両方に入っている40g/Lのセロビオースおよび50g/LのD−キシロースの混合物を用いて、SL01およびSL00の両方を培養した(図66)。振とうフラスコの培養において(図66a〜b)、SL01は、96時間において83%セロビオースを消費し、SL100(0.33g/L/時間〜0.46g/L/時間)と比べて41.2%高い平均のD−キシロース消費速度を示した。上昇した糖の消費速度と一致して、1.32倍に増した平均の生物量の増加速度が観察された(0.031gの乾燥細胞重量/L/時間〜0.072gの乾燥細胞重量/L/時間)。エタノール生産性は、0.07g/L/時間〜0.23g/L/時間であり、2.1倍を超えて増加した。48時間後に、糖1gにつき0.31gの最も高いエタノールの収率に達し、平均のエタノールの収率は糖1gにつき0.28gであり、SL00株と比べて23%の増加を示した。SL01の培養において、グルコースおよびD−キシロースの共発酵におけるグルコース抑制の特徴である遅延期を示すことなく、より速やかなD−キシロース消費速度が観察された。さらにまた、促進された生物量の増加およびエタノール産生が認められた。
バイオリアクターにおける混合糖の発酵のためにMultiforsシステム(Infors-HT, Bottmingen, Switzerland)を用いた。各容器は750mLの総容積を有していた。各容器に対して、pO2センサ、エアスパージャー、排気ガス冷却器、温度センサ、播種ポート、予備ポート、消泡センサ、pHセンサ、駆動軸、加熱器の区画、流量計測器、および蠕動ポンプシステムのセットを個々に備えつけた。全体のバイオリアクターシステムは、冷却システム、ThermoFlex900(Thermo Scientific, Waltham, MA)を備えている。
2%のグルコースを含んでいる2mLのSC−Ura−Leu培地において、酵母株の単一のコロニーをまず成長させ、それから250mLの振とうフラスコに入っている50mLの同じ培地に播種されて、混合糖の発酵試験にとって十分な細胞を得た。成長の1日後に、4%のセロビオースおよび5%のD−キシロース;4%のセロビオース、5%のキシロースおよび0.5%のグルコース;または4%のセロビオース、5%のキシロースおよび1%のグルコースを補ったYPA培地に、10mLの飽和した培養物を播種した。温度を30℃に維持させ、pHを、2規定のH2SO4または4規定のNaOHのいずれかの添加によって調節された5.5に維持させた。最初の48時間において、250rpmのインペラの速度を用いて空気の流量を0.5L/分に維持させた。その後に、空気の流量を0.2L/分に調節して、酸素制限条件下における高いエタノール産生を実現させた。3組のサンプルを種々の時点において採取し、OD600、糖濃度、およびエタノール濃度を上述のように決定した。
バイオリアクターの培養において(図66c〜d)、ほとんどすべてのセロビオースおよび66%のD−キシロースを48時間に消費し、44%の増加したD−キシロースの消費速度(0.47g/L/時間〜0.68g/L/時間)、および1.1倍に増加した生物量の増加速度(0.08gの乾燥細胞重量/L/時間〜0.17gの乾燥細胞重量/L/時間)を示した。エタノール生産性は、4.3倍(0.09g/L/時間〜0.50g/L/時間)を超えて増加し、エタノールの収率は糖1gにつき0.39gであった。振とうフラスコの培養と比べて、バッチ培養の開始時における低い細胞密度に起因して、最初の24時間における糖の消費速度が低くかった。
予想外にも、発酵にグルコースが加えられていなくとも、少量のグルコースが検出された(図66a〜b)。グルコース濃度は、約24時間後に振とうフラスコ(12.1g/L)およびバイオリアクター(17.5g/L)の両方において最大に達し、それから非常に低レベルまで低下した。SL00株においてグルコースが検出されなかったので、細胞外のグルコースは、解糖に使用されるグルコースの主な形態であるエピマーのα−グルコースへのβ−グルコースの緩やかな変換を生じ得る。典型的に、グルコースにおける相対的に低い濃度のために、β−グルコースは酵素的または化学的のいずれかにおいてα−グルコースに効率的に変換され得る(Bouffard et al., 1994)。しかし、改変されたSL01株において、β−グルコシダーゼによって触媒された過剰量のβ−グルコースは、細胞内においてセロビオースから生成され、わずかな部分が細胞外に分泌され得、これはβ−ガラクトースを用いた観察と同様である(Bouffard et al., 1994)。
また、工業的な条件においてリグノセルロース性の加水分解物には少量のグルコース(糖の総量の10%未満)が典型的に存在しているので、改変されたSL01株の発酵能力を、セロビオースD−キシロースおよびグルコースの混合物を用いて調べた。2つの濃度5g/Lまたは10g/Lのグルコースを、バイオリアクターにおける混合糖の炭素源として、40g/Lのセロビオースおよび50g/LのD−キシロースと組み合わせた。5g/Lのグルコースの場合(図67a〜b)、SL01によって、バッチ培養における48時間後に、81.7%のセロビオースが消費され、67.8%のD−キシロースが消費された。D−キシロースの消費速度は1.19倍(0.32g/L/時間〜0.69g/L/時間)まで上昇した。エタノール生産性は3.3倍(0.11g/L/時間〜0.46g/L/時間)まで増加し、エタノールの収率は糖1gにつき0.26g〜糖1gにつき0.33gであった。10g/Lのグルコースの場合(図67c〜d)、SL01によって、バッチ培養における48時間後に、83.8%のセロビオースが消費され、74.7%のD−キシロースが消費された。D−キシロースの消費速度は68%(0.45g/L/時間〜0.76g/L/時間)上昇した。エタノール生産性は2.1倍(0.16g/L/時間〜0.50g/L/時間)まで増加し、エタノールの収率は糖1gにつき0.30g〜糖1gにつき0.33gであった。予想通り、改変されたSL01株は、野生型のSL00株より、高効率の糖消費および高い割合のエタノール生産性の両方を示した。より重要なことに、糖の総量の10%までグルコースを有していてさえ、3つの糖の共発酵において顕著なグルコース抑制を示さず、これは、この手法が工業的な用途に発展させ得ることを示唆している。
3つのN. crassaのセロデキストリントランスポータNCU00801、NCU08114およびNCU00809を、β−グルコシダーゼNCU00130とともに導入したS. cerevisiae株D452−2において、同様の研究を実施した。細胞内β−グルコシダーゼ(NCU00130)を発現している形質転換体を、20g/リットルのセロビオースを含んでいるYSC培地おいて選択した。この作業に使用した株およびプラスミドは表17に記載されている(実施例12)。使用したプライマーを表28に示す。
20g/Lのグルコースまたは20g/Lのセロビオースを含んでいるYP培地において酵母を成長させて、それぞれキシロースまたはセロビオースの発酵実験のための播種物を調製した。20g/Lのグルコースまたはセロビオースを含んでいるYP培地から対数期にある細胞を回収し、滅菌水を用いて2回にわたって洗浄した後に播種した。250mLのフラスコに入っている40g/Lまたは80g/Lのキシロースを含んでいる50mLのYP培地を用いて、酸素制限条件下、1.0の初期のOD600、30℃において、すべてのフラスコ発酵実験を実施した。Sixfors Bioreactors(Appropriate Technical Resources, Inc)を用いて、適量の糖を含んでいる400mLのYP培地において、酸素の制限された250の条件下、200rpmの攪拌速度、30℃においてバイオリアクター発酵を実施した。初期の細胞密度をOD600=1.0に調節した。
UV-visible Spectrophotometer(Biomate 5, Thermo, NY)用いて、細胞の成長を600nmにおける吸光度(OD)によって観察した。Rezex ROA-Organic Acid H+(8%)カラム(Phenomenex Inc., Torrance, CA)を用いた、屈折率の検出器を備えている高速液体クロマトグラフィー(HPLC, Agilent Technologies 1200 Series)によって、グルコース、キシロース、キシリトール、グリセロール、酢酸塩およびエタノールの濃度を決定した。0.005規定のH2SO4を用いて、0.6mL/分の流速、50℃においてカラムを溶出した。
3つの形質転換体のすべては、セロビオースが単独の炭素源であるとき、成長可能であり、エタノールを産生可能であったが、3つの形質転換体は、セロビオースの異なる発酵速度(NCU00801>NCU08114>NCU00809)を示した。NCU00801およびNCU00130を発現している、最も早くセロビオースを発酵させる形質転換体(D801−130)は、4時間以内に40g/Lのセロビオースを消費し、16.8g/Lのエタノールを産生した。同一の培養条件下において、セロビオース発酵の容積測定の生産性(PEthanol/Cellobiose=0.7g/L/h)は、グルコース発酵の容積測定の生産性(PEthanol/Cellobiose=1.2g/L/h)より低く、セロビオースからのエタノールの収率(YEthanol/Cellobiose=0.42g/g)は、グルコースからのエタノールの収率(YEthanol/Cellobiose=0.43g/g)とほぼ同じであった。しかし、D801−130によるセロビオースの消費速度およびエタノールの収率は、β−グルコシダーゼの表面提示を介したセロビオースを発酵させるために改変したS. cerevisiae株を超えて改善が認められた(Kotaka et al., 2008; Nakamura et al., 2008)。これらの結果は、S. cerevisiaeにおけるNCU00801およびNCU00130の同時発現が効率的なセロビオース発酵をもたらすことを示唆している。
キシロースを効率的に発酵させるDA24−19株の開発の後に、セロデキストリントランスポータをコードしている遺伝子、およびβ−グルコシダーゼ酵素をコードしている遺伝子(NCU00801およびNCU00130)を、セロビオースおよびキシロースを同時に消費可能にさせる株に導入した。セロビオースの細胞内の加水分解のために、キシロース利用のグルコース抑制がこの株において緩和され得ると仮定された。NCU00801遺伝子をDA24−16のゲノムに組み込み、NCU00130を多コピーのプラスミドから発現させた。単独の炭素源としてセロビオースを含んでいる寒天プレートにおいて、生じた形質転換体DA24−18−BT3を選択した。
種々の量のセロビオースおよびキシロースを含んでいる培地において成長させたDA24−16−BT3株は、試験されたすべての条件において、セロビオースおよびキシロースを共発酵させ、0.38〜0.39g/gの収率を有してエタノールを産生した(図69)。3つの異なる条件:40g/Lのセロビオース、40g/Lのキシロースおよび40g/Lの両方の糖(合計80g/Lの糖)の下にDA24−16−BT3を培養することによって、共発酵の潜在的な相乗効果を試験した。驚くべきことに、DA24−16−BT3は、40g/Lのセロビオースまたは40g/Lのキシロースを別個に消費するために必要な期間と同じ期間内に、80g/Lのセロビオース/キシロースを共発酵させることができた(図70)。さらに、DA24−16−BT3は、単独の糖発酵(セロビオースまたはキシロース)からのエタノールの収率(0.31〜0.33g/g)と比べて、セロビオースおよびキシロースの混合物からより高い収率(0.39g/g)を有して、エタノールを産生した。また、エタノール生産性は、共発酵の間に0.27g/L/時間から0.65g/L/時間まで顕著に増加した。これらの結果は、セロビオースおよびキシロースの共発酵が全体的なエタノールの収率および生産性を向上させ得ることを証明した。また、この改変したS. cerevisiae株(DA24−16−BT3)を、セロビオースおよびキシロースを効率的に共発酵可能なP. stipitisと比較するために、発酵実験を行った。
エネルギーケインの組成物に基づく擬似の加水分解物(10g/Lのグルコース、80g/Lのセロビオース、40g/Lのキシロース)を使用した。異なるリグノセルロース性の植物の組成は広範な範囲において変わる。例えば、米国エネルギー省のバイオマスデータベースは、150のバイオマスサンプルの組成を一覧にしている(ウェブページeere.energy.gov/biomass/m/feedstock_databases.html)。これらのサンプルのセルロース対ヘミセルロースの割合は1.4〜19であり、その平均は2.3である。エネルギー農作物は、木質のバイオマスより高いヘミセルロースの含有量を典型的に有している。サトウキビのバガス、トウモロコシの茎および葉、モロコシのセルロース対ヘミセルロースの割合の平均は、それぞれ2.0、1.85および2.14である。したがって、われわれは、われわれの擬似の糖の実験計画において、2のグルカン/キシランの割合を使用した。改変した酵母は、生物燃料産生のためにβ−グルコシダーゼ活性について欠失している従来のセルラーゼカクテルとともに使用した。不完全なセルラーゼカクテルを用いた6〜30%の、グルコースへのグルカンの変換が報告されている(Medve et al., 1998)通り、バイオマスの加水分解過程は、リグノセルロース性の加水分解物において少量のグルコースを生じ得る。上述のすべての要因を考慮して、10g/Lのグルコース、80g/Lのセロビオースおよび40g/Lのキシロースの糖の組合せを擬似の糖の実験において選択した。
DA24−16BT3は、セロビオースおよびキシロースを急速に共発酵させる前にグルコースをまず消費した。わずかな播種物(OD600=1)を使用してさえ60時間以内に合計130g/Lの糖が消費された。対照的に、P. stipitisは、同一の培養条件における同じ期間内に糖の混合物の発酵を完了させ得なかった(図71)。DA24−16BT3は、60時間以内に48g/Lのエタノール(YEthanol/Sugars=0.37g/gおよびPEthanol/Sugars=0.79g/L/h)を産生した。
セロビオース消費の間の培地におけるセロデキストリンの一時的な蓄積を観察した(図72〜73)。蓄積したセロトリオースおよびセロテトラオースはセロビオースの枯渇後に同様に消費された。蓄積したセロデキストリンは、β−グルコシダーゼ(NCU00130)のグリコシル転移活性(Christakopoulos et al., 1994)によって生成され、両方向性(細胞内←→細胞外)のセロデキストリン輸送を容易にし得るセロデキストリントランスポータ(NCU00801)によって分泌されたと考えられる。蓄積したセロデキストリンが改変された酵母によって結果的に消費されるので、一時的なセロデキストリンのこの蓄積は収率をおそらく低下させない。しかし、セロトリオースおよびセロテトラオースの輸送率がセロビオースの輸送率より低下し得るので、それは生産性を低下させ得る。
少量のグルコースは共発酵の間に培地において常に検出された。少量でさえグルコースはキシロース発酵を抑制し得るので、グルコースのレベルを最小に維持する必要がある。セロデキストリントランスポータおよびβ−グルコシダーゼの相対的な発現レベルがグルコースの蓄積におそらく影響すると仮定された。この点を支持するように、NCU00801が酵母のゲノムに組み込まれている場合よりNCU00801が多コピーのプラスミドにおいて導入されている場合に、より多くのグルコースが培地に蓄積されることを観察した。多コピーのプラスミド上にNCU00801およびNCU00130の両方を含んでいる株(DA24−16BT3)は、DA24−16−BT3に認められるキシロース利用率より相対的に低いキシロース利用率を有しており、考えられる理由はグルコース抑制であった。セロデキストリントランスポータおよびβ−グルコシダーゼの発現レベルのさならる調節、または低下したグリコシル転移活性を有しているβ−グルコシダーゼの同定によって、共発酵の間におけるグルコースおよびセロデキストリンの蓄積を低下させることが可能であり得る。
また、2つの異なる酵母株:キシロースを発酵させるDA24−16株およびセロビオースを発酵させるDA452BTの混合培養によって、キシロースおよびセロビオースの共発酵を実現し得た(図75)。上述のように、酵母株DA24−16は、複数のキシロース利用酵素(野生型のキシロースリダクターゼ(XYL1)、変異体のキシロースリダクターゼ(mXYL1)、キシリトールデヒドロゲナーゼ(XYL2)、およびキシルロキナーゼ(XKS1))を発現していた。セロデキストリントランスポータNCU00801およびβ−グルコシダーゼ(NCU00130)を発現させるためにD452を改変することによって、D452BTを形成した。混合培養において、DA24−16株はキシロースを取込み(図75aにおいてキシロース分子は緑の五角形として示されている)、酵素XYL1(野生型および変異体)、XYL2およびXYL3を用いてキシロースを代謝させ、他の株D452BTは、トランスポータNCU00130を用いてセロビオースを取込み可能であり(図75aにおいてセロビオース分子は2つの赤の六角形として示されている)、酵素NCU00130を用いてセロビオースをグルコースに変換させた。したがって、混合培養は、キシロースおよびセロビオースの両方を共発酵させて、エタノールを産生可能であった(図75b)。
本研究において、新規な方法によってS. cerevisiaeによるヘキソース糖およびペントース糖の共発酵が可能なことを証明した。キシロース利用経路をセロデキストリン輸送系と組み合わせることによって、グルコース抑制が引き起こす問題を解消させた。結果として、改変された酵母は、セルロース性の加水分解物における代謝不可能な2つの糖をエタノールへと相乗的に共発酵させた。本明細書に記載の新規な共発酵方法は、糖化および発酵の両方に関するリグノセルロースに関する手法を進歩させる。最も伝統的な真菌のセルラーゼカクテルは、β−グルコシダーゼについて不完全であり、酵母によって効率的に発酵されないセロビオースをともなうセルロース加水分解を停止させる。結果として、セロビオースをグルコースに変換するためには、過剰のβ−グルコシダーゼを加える必要がある。セロビオース/キシロースを共発酵させる酵母によって、わずかなβ−グルコシダーゼ活性を有しているこれらのセルラーゼカクテルを使用すること、酵素の使用量の低下、およびセルロースの糖化処理に関する費用の低下が可能になる。さらに、セロビオースおよびキシロースの共発酵の間における相乗効果は、エタノールの生産性を有意に高め、したがって発酵の経済的側面を改善させる。リグノセルロース性材料の前処理および加水分解に由来する少量のグルコースの存在は、改変された酵母の能力に影響を与えずに、ヘキソース糖およびペントース糖の混合物をエタノールに変換させる。
本研究は、植物のバイオマスから得られた加水分解物を模倣するように意図されている種々の糖の混合物を発酵させる、改変されたS. cerevisiaeの能力を評価することを含んでいた。セロデキストリンおよびキシロースを共発酵させるこの株の能力は、前処理された植物のバイオマスの同時の糖化および共発酵(SSCF)において特に有用である。SSCFの間において、ヘミセルロースは、酸の前処理によってまず加水分解され、キシロースおよび未だ結晶性のセルロースを生じる。それから、真菌のセルラーゼおよび本明細書に記載の酵母株が加えられ、キシロースおよびセロビオースをともにエタノールに変換可能になる。この手順における細胞外グルコースのわずかな産生のために、キシロース利用の抑制が低減され、共発酵は速やかかつ相乗的に進行する。
本研究に使用されたS. cerevisiae株は研究室株であるが、改変された株の発酵の性能は、発表済の結果と比べて非常に目覚しい。重要な発酵パラメータ(収率および生産性)は、プラットフォームとして産業用株を使用することによってさらに向上されている。この共発酵の方法の用途はエタノール産生に限定されない。これは基礎的な技術であるため、本明細書に示されている方法は、任意の他の製品多様化技術と組み合わせて、商品としての化学物質および先進的な生物燃料を生成し得る。
〔実施例18:キシランに基づいて成長させたN. crassaのトランスクリプトーム分析〕
リグノセルロースのバイオマスは、セルロース、ヘミセルロースおよびリグニンによって構成されている。実施例1〜3には、N. crassaのトランスクリプトームおよびセクレトームの分析を介した、セルロースに基づく成長にとって重要な遺伝子の発見について説明されている。本実施例では、遺伝子がヘミセルロースの利用に重要であることを決定するために、キシランに基づく成長の間における、N. crassaゲノムの発現プロファイルを試験した。
リグノセルロースのバイオマスは、セルロース、ヘミセルロースおよびリグニンによって構成されている。実施例1〜3には、N. crassaのトランスクリプトームおよびセクレトームの分析を介した、セルロースに基づく成長にとって重要な遺伝子の発見について説明されている。本実施例では、遺伝子がヘミセルロースの利用に重要であることを決定するために、キシランに基づく成長の間における、N. crassaゲノムの発現プロファイルを試験した。
100mLの1倍のVogel's塩の最少培地(2%のスクロース)に対してWTまたはΔxlnR株の10日齢の分生子を、106の分生子/mLにおいて播種し、16時間にわたって恒明下の25℃において成長させ、1倍のVogel'sのみの培地を用いて洗浄した。それから、培地において2%のスクロースまたは2%のブナ材のキシランを単独の炭素源として有している1倍のVogel's塩に分生子を移し、4時間にわたって放置して成長させた。ろ過によって菌糸を回収し、液体窒素において直ちに瞬間冷凍した。製造者の指示にしたがってTRIzol(Invitrogen)を用いて、総RNAを単離し、DNase(Turbo DNA‐free kit; Ambion)(Kasuga, Townsend et al., 2005)を用いて処理した。
cDNA合成および標識のために、使用した総RNAがサンプルごとに10μgであった点を除いて、製造者の仕様書にしたがってProntoキット(カタログNo.40076;Corning)を使用した。
上述のようにマイクロアレイハイブリダイゼーションおよびデータ分析を行った(Tian, Kasuga et al., 2007)。画像の取得のためにGenePix 4000Bスキャナ(Axon Instruments)を使用し、ハイブリダイゼーションシグナルの定量化および未加工データの収集にGenePix Pro6ソフトウェアを使用した。BAGEL(Bayesian analysis of gene expression levels)ソフトウェアプログラム(Townsend and Hartl 2002; Townsend 2004)を用いることによって、標準化した発現値を分析した。354の遺伝子は、キシランに基づいて成長させたN. crassaにおいて2倍を超えて誘導されていることが分かった。図76にその一覧を示す。
〔実施例19:キシランに基づいて成長させたN. crassaのセクレトーム分析〕
ショットガンプロテオミクス(shotgun proteomics)手法を用いて、キシランに基づく成長の間におけるN. crassaのセクレトームを分析した。キシラン培養物から得られた上清を、トリプシン処理し、タンデムの液体クロマトグラフィーナノエレクトロスプレーイオン化質量分析によって分析した。
ショットガンプロテオミクス(shotgun proteomics)手法を用いて、キシランに基づく成長の間におけるN. crassaのセクレトームを分析した。キシラン培養物から得られた上清を、トリプシン処理し、タンデムの液体クロマトグラフィーナノエレクトロスプレーイオン化質量分析によって分析した。
質量分析のサンプルを以下のように調製した。N. crassaの野生株を、2%のキシランの培地において4または7日にわたって成長させた。培養上清を遠心分離によって単離し、0.22μmのフィルタに通してろ過し、MWCO PESスピン濃縮器を用いて10倍に濃縮させた。それから、3.36mgの尿素、5μLの1MのTris pH8.5および5μLの100mMのDTTを、100μLの濃縮された培養上清に加え、混合物を1時間にわたって60℃において加熱した。加熱後、700μLの25mMのジカルボン酸アンモニウムおよび140μLのメタノールを溶液に加え、その後に50mMの酢酸ナトリウム pH5.0における50μLの100μg/mLのトリプシンを用いて処理した。トリプシンを残したまま、基底のpHにおいて8〜9時間にわたって転倒させながら、37℃において一晩にわたって反応させた。消化の後に、スピードバックによって乾燥するまで容積を減らし、300μlのMilliQ水を用いて3回にわたって洗浄した。最終容積は100μlであった。製造者の指示にしたがって、OMIX微量抽出ピペットチップを用いることによって、サンプルにおける残余の塩を除去した。アセトニトリルを蒸発によって除去した。サンプル溶液は、0.1%〜1%のTFAを有している水性溶液であり、最終容積は10マイクロリットルを超えていた。
〔実施例20:分泌タンパク質をコードすると予測されているキシラン誘導遺伝子の分析〕
トランスクリプトームおよびセクレトームの分析結果は、合計71の遺伝子を示し、このうち55の遺伝子が分泌されていた。これらの遺伝子の一覧を表29に示す。欠失株は69遺伝子のうち46遺伝子について入手可能であった。これらの46遺伝子のうちの6つの株は異核共存体であり、したがって、分泌タンパク質の総量、キシロースの存在量およびアゾ−エンド−キシラナーゼ活性について、残りの40の株を分析した。結果を図77に示す。
トランスクリプトームおよびセクレトームの分析結果は、合計71の遺伝子を示し、このうち55の遺伝子が分泌されていた。これらの遺伝子の一覧を表29に示す。欠失株は69遺伝子のうち46遺伝子について入手可能であった。これらの46遺伝子のうちの6つの株は異核共存体であり、したがって、分泌タンパク質の総量、キシロースの存在量およびアゾ−エンド−キシラナーゼ活性について、残りの40の株を分析した。結果を図77に示す。
サンプルを以下のように調製した。2%のキシランのVogel's培地の100mLにおいて、106の分生子/mLの10日齢の分生子を成長させた。各株について2組を調製した。培養物を遠心分離によって単離し、アッセイに使用した。
Bradfordのタンパク質濃度を測定して、分泌タンパク質の総量を決定した。BSA標準物:0μg/mL、50μg/mL、100μg/mL、250μg/mL、および500μg/mLを用いてストックを調製した。複数のチャネルのあるピペットを使用して、200μLのBradford溶液を96ウェルのプレートに移した。10μLのサンプルおよび10μLの標準物を10分にわたって室温においてインキュベートした。595nmにおける吸光度を読み取り、タンパク質濃度を決定した。
キシロースを測定するために利用したアッセイは、Bailey et al., 1992(J Biotech 23: 257-270)から変更した。キシロース標準物をH2Oにおいて調製した。濃縮された0.8Mのキシロース(10mLに1.2g)について、標準物は、0mM、8mM(1:100希釈;990μl+10μl)、20mM(1:100希釈;975μl+25μl)、40mM(1:100希釈;950μl+50μl)、80mM(1:100希釈;900μl+100μl)、および160mM(1:100希釈;800μl+200μl)を含んでいた。複数のチャネルのあるピペットを使用して、深いウェルの96ウェルプレートに900μLの基質溶液を加えた。100μLの培養上清および標準物を加え、放置して5分にわたって50℃においてインキュベートした。サンプルを3400rpmにおいて10分にわたって遠心分離した。複数のチャネルのあるピペットを使用して96ウェルのPCRプレートに75μLのDNS溶液を加えた。5μLの溶液を反応物から取り出し、DNS溶液の入っているPCRプレートに加えた。サンプルの冷却後に、それらを透明な平底プレートに移し、540nmにおける吸光度を読み取った。基質溶液(500mL)は、ブナ材のキシラン(5g;10mg/mL)、3MのNaOAc、pH5.0(8.33mL;50mM)、水(491mL)を含んでおり、20分にわたって乾熱滅菌された。DNS溶液(100mL)は、3,5−ジニトロサリチル酸(707mg)、Rochelle塩(Na Kの酒石酸塩)(20.4g)、メタ重亜硫酸ナトリウム(553mg)、フェノール(507μL)、および水(94.4mL)を含んでいた。
Megazymeが提供しているキットを用いてアゾ−エンド−キシラナーゼ活性を測定した。このアッセイは、色素と複合化させたキシラン鎖から遊離した色素の量を分光光度に基づいて測定することによって、サンプルにおけるエンド−キシラナーゼ活性の程度を直接的に測定する。存在している酵素が多いほど、多くの色素が放出される。別々のFalconチューブに入っている450μLの酢酸ナトリウム緩衝液(50mM、pH4.5)に50μLの上清を加えることによって、すべての上清サンプルを希釈した。次に、Falconチューブを10分にわたって予熱した。基質溶液をすべてのサンプル(500μL/サンプル)についてチューブに加えた。サンプルおよび基質溶液を10分にわたって40℃の水槽にいれ、あらかじめそれらを平衡化させた。500μLの基質溶液を1:10に希釈したサンプルに加え、10秒にわたってボルテックスにかけ、10分にわたって40℃においてインキュベートした。各サンプルに2.5mLの沈降溶液(95%のエタノール)を加えることによって反応を停止させ、10秒にわたってボルテックスにかけた。チューブを10分にわたって室温において静置した。チューブを10秒にわたってボルテックスにかけ、それから10分にわたって1000rpmおよび室温において遠心分離にかけた。各チューブからの1mLの上清溶液をキュベットへ直接に入れ、590nmにおける吸光度を測定した。この手順のために使用したブランクは2.5mLの沈降物溶液に加えられた500μLの基質溶液から得られた上清であった。
結論として、N. crassaにおけるヘミセルロースの分解に影響を与えると、ここで同定された遺伝子の調節は、植物の細胞壁を崩壊させる向上した能力、および植物の細胞壁の構成要素(例えばヘミセルロース)に基づく促進された成長を有している株の作製を容易にすると予想される。興味深い遺伝子としては、推定のグルカミラーゼをコードしているNCU05157;推定のアラビノフラノシダーゼをコードしているNCU02343;保存されている推定のタンパク質をコードしているNCU05137;推定のアセチルキシランエステラーゼ前駆体をコードしているNCU05159;保存されている機能未知のタンパク質をコードしているNCU09133;および機能未知のタンパク質をコードしているNCU10040が挙げられる。
ヘミセルロースに基づく細胞の成長は、NCU01517、NCU09133またはNCU10040によってコードされているポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備することによって促進される。上記宿主細胞は、上記組換えポリヌクレオチドが発現されるような、ヘミセルロースを含んでいる培地において培養される。上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。
〔実施例21:ΔNCU05137株のさらなる分析〕
実施例1〜3および18〜20に記載のように、NCU05137は、Miscanthus、Avicelまたはキシランのうちのいずれかに基づくN. crassaの成長の間に過剰発現された推定の分泌タンパク質である。Avicelに基づいて成長させた、NCU05137を欠いているN. crassaの欠失株は、エンドグルカナーゼ、β−グルコシダーゼおよびAvicelaseの増大した活性を示した。キシランに基づいて成長させたNCU05137欠失株は、アゾ−エンド−キシラナーゼの増大した活性を示した。本実施例に述べられるように、ΔNCU05137株に認められた表現型がNCU05137遺伝子の消失に起因することを確認するために、ΔNCU05137の補完を行った。
実施例1〜3および18〜20に記載のように、NCU05137は、Miscanthus、Avicelまたはキシランのうちのいずれかに基づくN. crassaの成長の間に過剰発現された推定の分泌タンパク質である。Avicelに基づいて成長させた、NCU05137を欠いているN. crassaの欠失株は、エンドグルカナーゼ、β−グルコシダーゼおよびAvicelaseの増大した活性を示した。キシランに基づいて成長させたNCU05137欠失株は、アゾ−エンド−キシラナーゼの増大した活性を示した。本実施例に述べられるように、ΔNCU05137株に認められた表現型がNCU05137遺伝子の消失に起因することを確認するために、ΔNCU05137の補完を行った。
ccgIプロモータの制御下にあるC末端にGFPタグを有しているNCU05137を含んでいるプラスミドを生成した。NCU05137−GFPコンストラクトを用いてN. crassaの分生子を形質転換した。Neurosporaの手順(ウェブページfgsc.net/Neurospora/NeurosporaProtocolGuide.htm)にしたがって実験を行った。
野生型、ΔNCU05137、ΔNCU05137−NCU05137−GFP株の総分泌タンパク質およびカルボキシルメチルセルロース(CMC)活性を測定した。各株の培養物から得られた100μLの上清を取り、当該上清を900μLのBradford Dyeに加え、595nmにおける吸光度を測定することによって、総分泌タンパク質を測定した。各株の培養物から得られた上清の20倍希釈物およびCMCキット(Megazyme SCMCL)を用いて、アゾ−CMC活性を測定した。ΔNCU05137ノックアウト株は分泌タンパク質およびCMC活性の増大したレベルを示した。ΔNCU05137株へのGFPタグつきのNCU05137の導入は、これらのレベルを低下させ、野生型のレベルに戻した(図78)。
さらに、補完された株におけるNCU05137−GFPの局在を観察した。NCU05137−GFPは、分生子の細胞壁、菌糸の先端に局在した(図79〜80)。これらのデータは、GFPタグつきのNCU05137タンパク質が、十分に機能的であり、精製およびこのタンパク質の生化学的活性を扱う実験に使用され得ることを示している。
したがって、NCU05137の通常の機能は、セルラーゼ遺伝子およびヘミセルラーゼ遺伝子の発現の誘導と関連付けられているシグナリング過程を阻害することであり得る。細胞におけるNCU05137またはNCU05137相同物の発現の低下は、当該細胞におけるセルラーゼ活性およびヘミセルラーゼ活性を低下させ、続いてセルロースまたはヘミセルロースに基づく当該細胞の成長を抑制すると考えられる。セルロースまたはヘミセルロースに基づく細胞の成長は、NCU05137によってコードされているポリペプチドをコードしている外因性のポリヌクレオチドを含んでいる宿主細胞を準備することによって、促進される。上記外因性のポリヌクレオチドの発現が抑制され、上記細胞はセルロースまたはヘミセルロースを含んでいる培地において培養される。上記宿主細胞細胞は、上記外因性のポリヌクレオチドの発現が抑制されていない細胞と比べて、上記培地においてより早く成長する。
〔実施例22:NCU07705のさらなる分析〕
NCU07705の発現は、セルロースに基づくN. crassaの成長の間に上方制御されることがわかった。NCU07750によってコードされているポリペプチドのBLAST分析によって、当該ポリペプチドは、C6ジンクフィンガードメインを含んでいる多くの転写因子と高い類似性を有していることが明らかになった(図1)。セルロースの利用におけるNCU07705の役割をさらに調べるために、NCU07705を欠いている欠失株の表現型を評価した。
NCU07705の発現は、セルロースに基づくN. crassaの成長の間に上方制御されることがわかった。NCU07750によってコードされているポリペプチドのBLAST分析によって、当該ポリペプチドは、C6ジンクフィンガードメインを含んでいる多くの転写因子と高い類似性を有していることが明らかになった(図1)。セルロースの利用におけるNCU07705の役割をさらに調べるために、NCU07705を欠いている欠失株の表現型を評価した。
ΔNCU07705株は、単独の炭素源としての2%のセルロース(Avicel)、PASCまたはCMCに基づいて成長できなかった(表30)が、スクロース、キシランおよびキシロースに基づいて野生型の株と同様の動態を示して成長した。NCU07705がセルラーゼの発現の制御に役割を果たしているか否かを決定するために、セルロースに基づくΔNCU07705の成長の間におけるセルラーゼ遺伝子およびヘミセルラーゼ遺伝子の発現が試験された。野生型の株(FGSC2489)およびΔNCU0775株から得られた10日齢の分生子をVogel'sの液体MM(Vogel 1956)に播種し、16時間にわたって成長させた。菌糸を遠心分離し、1倍のVogel's塩を用いて洗浄し、それから2%のスクロースまたは2%のAvicelを含んでいるVogel's培地のいずれかに移し、4時間にわたって恒明下において成長させた。それらをろ過によって回収し、液体窒素において直ちに凍結させた。製造者の指示にしたがってTRIzol(Invitrogen, Carlsbad, CA)を用いて総RNAを単離し、DNase(Turbo DNA-free kit, Ambion/Applied Biosystems, Foster City, CA)を用いて処理した(Kasuga et al., 2005)。ChipShot(商標)Indirect Labeling/Clean-Up System(カタログNo.Z4000、Promega, Madison, WI)およびCyDye Post-Labeling Reactive Dye Pack(カタログNo.RPN5661、GE Healthcare, Piscataway, NJ)を使用して、RNAの使用量が10μgであることを除いて、製造者の指示にしたがってcDNAを合成し、標識した。製造者の仕様書にしたがって、マイクロアレイハイブリダイゼーションのために、Pronto! Hybridization Kit(カタログNo.40076、Corning, Lowell, MA)を使用した。
上述のようにデータ分析を行った(Tian et al., 2007)。GenePix 4000Bスキャナ(Axon Instruments, Union City, CA)を使用して、画像を取得し、GenePix Pro6ソフトウェアを使用して、ハイブリダイゼーションシグナルを定量化し、未加工のデータを収集した。BAGEL(Bayesian Analysis of Gene Expression Levels)を用いることによって、標準化された発現値を分析した(Townsend and Hartl, 2002)。推定のセルラーゼ遺伝子はΔNCU07705において誘導されないにもかかわらず、推定のヘミセルラーゼ遺伝子の誘導は影響されなかった(以下の表30を参照)。したがって、NCU07705は、cdr−1(cellulose degradation regulator 1)と名づけられている。
したがって、セルロースに基づく細胞の成長は、NCU07705によってコードされているポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備することによって、促進される。上記宿主細胞は、上記組換えポリヌクレオチドが発現されるような、セルロースを含んでいる培地において培養される。上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する。
〔参考文献〕
Arendt, C. S., K. Ri, et al. (2007). “Genetic selection for a highly functional cysteine-less membrane protein using site saturation mutagenesis.” Anal Biochem 365(2):185-193.
Bai, F. W., W. A. Anderson, and M. Moo-Young (2008). “Ethanol fermentation technologies from sugar and starch feedstocks.” Biotechnol Adv 26(1): 89-105.
Bailey, M. J., P. Biely, and K. Poutanen (1992). “Interlaboratory testing of methods for assay of xylanase activity.” J Biotechnol 23:257-270.
Bailey, J. E. and D. F. Ollis (1986). Biochemical Engineering Fundamentals, McGraw-Hill Book Company, NY.
Basso, L. C., H. V. de Amorim, et al. (2008). “Yeast selection for fuel ethanol production in Brazil.” FEMS Yeast Res 8(7):1155-1163.
Becker, D. M. and V. Lundblad (2001). “Introduction of DNA into yeast cells.” Curr Protoc Mol Biol Chapter 13, Unit13.7.
Bendtsen, J. D., H. Nielsen, et al. (2004). “Improved prediction of signal peptides: SignalP 3.0.” J. Mol. Biol 340(4):783-95.
Bengsston, O., B. Hahn-Hagerdal, and M. F. Gorwa-Grauslund (2009). “Xylose reductase from Pichia stipitis with altered coenzyme preference improves ethanolic xylose fermentation by recombinant Saccharomyces cerevisiae.” Biotechnol Biofuels 2:9.
Bhat, K. M. and R. Maheshwari (1987). “Sporotrichum thermophile Growth, Cellulose Degradation, and Cellulase Activity.” Appl Environ Microbiol 53(9):2175-2182.
Bouffard, G. G., K. E. Rudd, and S. L. Adhya (1994). “Dependence of lactose metabolism upon mutarotase encoded in the gal operon in Escherichia coli.” J Mol Biol 244(3):269-278.
Bouws, H, A. Wattenberg and H. Zorn (2008). “Fungal secretomes--nature's toolbox for white biotechnology.” Appl Microbiol Biotechnol 80(3):381-388.
Brat, D., E. Boles, and B. Wiedemann (2009). “Functional Expression of a Bacterial Xylose Isomerase in Saccharomyces cerevisiae.” Appl Environ Microbiol 75(8):2304-2311.
Canevascini, G. (1988). “Cellobiose dehydrogenase from Sporotrichum thermophile.” Method Enzymol 160:443-448.
Cantarel, B. L., P. M. Coutinho, et al. (2009). “The Carbohydrate-Active EnZymes database (CAZy): an expert resource for glycogenomics.” Nucleic Acids Res 37(Database issue):D233-238.
Chauve, M., H. Mathis, et al. (2010). “Comparative kinetic analysis of two fungal beta-glucosidases.” Biotechnol Biofuels 3(1):3.
Christakopoulos, P., M. K. Bhat, et al. (1994). “Enzymatic synthesis of trisaccharides and alkyl beta-D-glucosides by the transglycosylation reaction of beta-glucosidase from Fusarium oxysporum.” Int J Biol Macromol 16(6):331-334.
Christianson, T.W., R. S. Sikorskim, et al., (1992). “Multifunctional yeast high-copy number shuttle vectors.” Gene, 110(1):119-122.
Chu, B. C. and H. Lee (2007). “Genetic improvement of Saccharomyces cerevisiae for xylose fermentation.” Biotechnol Adv 25(5):425-441.
Colot, H. V., G. Park, et al. (2006). “A high-throughput gene knockout procedure for Neurospora reveals functions for multiple transcription factors.” Proc Natl Acad Sci USA 103(27):10352-10357.
Cullen, D., L. J. Wilson, et al. (1987). “Sequence and centromere proximal location of a transformation enhancing fragment ans1 from Aspergillus nidulans.” Nucleic Acids Res 15(22):9163-9175.
Davies, G. J., A. M. Brzozowski, et al. (2000).”Structure and function of Humicola insolens family 6 cellulases: structure of the endoglucanase, Cel6B, at 1.6 A resolution.” Biochem J 348 Pt 1:201-207.
Davis, R. H. (2000). “Neurospora: Contributions of a model organism.” New York, Oxford UniversityPress.
Davis, R. H. and D. D. Perkins (2002). “Timeline: Neurospora: a model of model microbes.” Nat Rev Genet 3(5):397-403.
de Groot, M. J., W. Prathumpai, et al. (2005). “Metabolic control analysis of Aspergillus niger L-arabinose catabolism.” Biotechnol Prog 21(6):1610-1616.
Dementhon K, G. Iyer, and N. L. Glass (2006). “VIB-1 is required for expression of genes necessary for programmed cell death in Neurospora crassa.” Eukaryot Cell 5(12):2161-2173.
Doran-Peterson, J., A. Jangid, et al. (2009). “Simultaneous saccharification and fermentation and partial saccharification and co-fermentation of lignocellulosic biomass for ethanol production.” Methods Mol Biol 581:263-280.
Drissen, R. E. T., R. H. W. Mass, et al. (2009). “Modelling ethanol production from cellulose: separate hydrolysis and fermentation versus simultaneous saccharification and fermentation.” Biocatal Biotransfor 27(1):27-35.
Dunlap, J. C., K. A. Borkovich, et al. (2007). “Enabling a Community to Dissect an Organism: Overview of the Neurospora Functional Genomics Project.” Adv Genet 57:49-96.
du Preez, J. C., M. Bosch, and B. A. Prior (1986). “The fermentation of hexose and
pentose sugars by Candida shehatae and Pichia stipitis.” Appl Microbiol Biotechnol 23(3):228-233.
Eberhart, B. M., R. S. Beck, et al. (1977). “Cellulase of Neurospora crassa.” J Bacteriol 130(1):181-186.
Ellis, K. J. and J. F. Morrison (1982). “Buffers of constant ionic strength for studying pH-dependent processes.” Methods Enzymol 87:405-426.
Espagne, E., O. Lespinet, et al. (2008). “The genome sequence of the model ascomycete fungus Podospora anserina.” Genome Biol 9(5):R77.
Fonseca, C. R. Romao, et al. (2007). “L-Arabinose transport and catabolism in yeast.” FEBS J 274(14):3589-3600.
Freer, S. N. (1991). “Fermentation and aerobic metabolism of cellodextrins by yeasts.” Appl Environ Microbiol 57(3):655-659.
Freer, S. N. and R. V. Greene (1990). “Transport of glucose and cellobiose by Candida wickerhamii and Clavispora lusitaniae.” J Biol Chem 265(22):12864-12868.
Galagan, J. E., S. E. Calvo, et al. (2003). “The genome sequence of the filamentous fungus Neurospora crassa.” Nature 422(6934):859-868.
Gems, D., I.L. Johnstone, and A. J. Clutterbuck (1991). “An autonomously replicating plasmid transforms Aspergillus nidulans at high frequency.” Gene 98(1):61-67.
Guindon, S. and O. Gascuel (2003). “A simple, fast, and accurate algorithm to estimate large phylogenies by maximum likelihood.” Syst Biol 52(5):696-704.
Hahn-Hagerdal, B., K. Karhumaa, et al. (2007). “Towards industrial pentose-fermenting yeast strains.” Appl Microbiol Biotechnol 74(5):937-953.
Hector, R. E., N. Qureshi et al. (2008). “Expression of a heterologous xylose transporter in a Saccharomyces cerevisiae strain engineered to utilize xylose improves aerobic xylose consumption.” Appl Microbiol Biotechnol 80(4):675-684.
Himmel, M. E., S. Y. Ding, et al. (2007). “Biomass recalcitrance: engineering plants and enzymes for biofuels production.” Science 315(5813):804-807.
Hosaka, K., J. Nikawa, et al., (1992). “A dominant mutation that alters the regulation of INO1 expression in Saccharomyces cerevisiae.” J Biochem 111(3):352-358.
Jacobson, D. J., A. J. Powell, et al. (2004). “Neurospora in temperate forests of western North America.” Mycologia 96(1):66-74.
Jeffries, T. W., I.V. Grigoriev, et al. (2007). “Genome sequence of the lignocellulose-bioconverting and xylose-fermenting yeast Pichia stipitis.” Nat Biotechnol 25(3):319-326.
Kasuga, T. and N. L. Glass (2008). “Dissecting colony development of Neurospora crassa using mRNA profiling and comparative genomics approaches.” Eukaryot Cell 7(9): 1549-64.
Kasuga, T., J. P. Townsend, et al. (2005). “Long-oligomer microarray profiling in Neurospora crassa reveals the transcriptional program underlying biochemical and physiological events of conidial germination.” Nucleic Acids Res 33(20): 6469-85.
Katahira, S., M. Ito, et al. (2008). “Improvement of ethanol productivity during xylose and glucose co-fermentation by xylose-assimilating S. cerevisiae via expression of glucose transporter Sut1.” Enzyme Microb Tech 43(2):115-119.
Katahira, S., A. Mizuike, et al. (2006). “Ethanol fermentation from lignocellulosic hydrolysates by a recombinant xylose- and cellooligosaccharide-assimilating yeast strain.” Appl Microbiol Biotechnol 72(6):1136-1143.
Korkhin, Y., A. J. Kalb(Gilboa), et al. (1998). “NADP-dependent bacterial alcohol dehydrogenases: crystal structure, cofactor-binding and cofactor specificity of the ADHs of Clostridium beijerinckii and Thermoanaerobacter brockii.” J Mol Biol 278(5):967-981.
Kotaka, A., H. Bando, et al. (2008). “Direct ethanol production from barley beta-glucan by sake yeast displaying Aspergillus oryzae beta-glucosidase and endoglucanases.” J Biosci Bioeng 105(6):622-627.
Kotter, P. and M. Ciriacy (1993). “Xylose fermentation by Saccharomyces cerevisiae.” Appl Microbiol Biotechnol 38(6):776-783.
Kubicek, C. P., R. Messner, et al. (1993). “Triggering of cellulase biosynthesis by cellulose in Trichoderma reesei. Involvement of a constitutive, sophorose-inducible, glucose-inhibited beta-diglucoside permease.” J Biol Chem 268(26):19364-19368.
Kumar, R., S. Singh, and O. V. Singh (2008). “Bioconversion of lignocellulosic biomass: biochemical and molecular perspectives.” J IndMicrobiol Biotechnol 35(5):377-391.
Kuyper, M, H. R. Harhangi, et al. (2003). “High-level functional expression of a fungal xylose isomerase: the key to efficient ethanolic fermentation of xylose by Saccharomyces cerevisiae? FEMS Yeast Res 4(1):69-78.
Lang, J. M. and V. P. Cirillo (1987). “Glucose transport in a kinaseless Saccharomyces cerevisiae mutant.” J Bacteriol 169(7):2932-2937.
Leandro, M. J., P. Gonsalves, and I. Spencer-Martins (2006). “Two glucose/xylose transporter genes from the yeast Candida intermedia: first molecular characterization of a yeast xylose-H+ symporter.” Biochem J 395(3):543-549.
Linder, M. and T. T. Teeri (1996). “The cellulose-binding domain of the major cellobiohydrolase of Trichoderma reesei exhibits true reversibility and a high exchange rate on crystalline cellulose.” Proc Natl Acad Sci USA93(22):12251-12255.
Lynd, L. R., M. S. Laser, et al. (2008). “How biotech can transform biofuels.” Nat. Biotechnol. 26(2):169-172.
Lynd, L. R., P. J. Weimer, et al. (2002). “Microbial cellulose utilization: fundamentals and biotechnology.” Microbiol Mol Biol Rev 66(3):506-77, table of contents.
Madhavan, A., S. Tamalampudi, et al. (2009). “Xylose isomerase from polycentric fungus orpinomyces: gene sequencing, cloning, and expression in Saccharomyces cerevisiae for bioconversion of xylose to ethanol.” Appl Microbiol Biotechnol 82(6):1067-1078.
Martin, F., A. Kohler, et al. (2010). “Perigord black truffle genome uncovers evolutionary origins and mechanisms of symbiosis.” Nature 464(7291):1033-1038.
Martinez, D., R. M. Berka, et al. (2008). “Genome sequencing and analysis of the biomass-degrading fungus Trichoderma reesei (syn. Hypocrea jecorina).” Nat Biotechnol 26(5):553-60.
Martinez, D., J. Callacombe, et al. (2009). “Genome, transcriptome, and secretome analysis of wood decay fungus Postia placenta supports unique mechanisms of lignocellulose conversion.” Proc Natl Acad Sci USA106(6):1954-1959.
Martinez, D., L. F. Larrondo, et al. (2004). “Genome sequence of the lignocellulose degrading fungus Phanerochaete chrysosporium strain RP78.” Nat Biotechnol 22(6):695-700.
Matsushika, A., S. Watanabe, et al. (2008). “Expression of protein engineered NADP+-dependent xylitol dehydrogenase increases ethanol production from xylose in recombinant Saccharomyces cerevisiae.” Appl Microbiol Biotechnol 81(2):243-255.
Matteucci, M. D. and M. H. Caruthers (1980). Tetrahedron Lett 21:719-722.
McCluskey, K. (2003). “The Fungal Genetics Stock Center: from molds to molecules.” Adv Appl Microbiol 52:245-262.
Medve, J., J. Karlsson, et al. (1998). “Hydrolysis of microcrystalline cellulose by cellobiohydrolase I and endoglucanase II from Trichoderma reesei: adsorption, sugar production pattern, and synergism of the enzymes.” Biotechnol Bioeng 59(5):621-34.
Miyasaka, H. (1999). “The positive relationship between codon usage bias and translation initiation AUG context in Saccharomyces cerevisiae.” Yeast 15(8):633-637.
Nair, N. and H. Zhao (2007). “Biochemical characterization of an L-Xylulose reductase from Neurospora crassa.” Appl Environ Microbiol 73(6):2001-2004.
Nakamura, N., R. Yamada, et al. (2008). “Effective xylose/cellobiose co-fermentation and ethanol production by xylose-assimilating S. cerevisiae via expression of β-glucosidase on its cell surface.” Enzyme Microb Tech 43(3):233-236.
Noguchi, Y., M. Sano, et al. (2009). “Genes regulated by AoXlnR, the xylanolytic and cellulolytic transcriptional regulator, in Aspergillus oryzae.” Appl Microbiol Biotechnol 85(1):141-154.
Notredame, C., D. G. Higgins, and J. Heringa (2000). “T-Coffee: A novel method for fast and accurate multiple sequence alignment.” J Mol Biol 302(1):205-217.
Pandit, A. and R. Maheshwari (1996). “Life-history of Neurospora intermedia in a sugar cane field.” J Biosci (Bangalore) 21(1):57-79.
Pauly, T. A., J. L. Ekstrom, et al. (2003). “X-ray crystallographic and kinetic studies of human sorbitol dehydrogenase.” Structure 11(9):1071-1085.
Pedelacq, J. D., S. Cabantous, et al. (2006). “Engineering and characterization of a superfolder green fluorescent protein.” Nat Biotechnol 24(1):79-88.
Perkins, D. D., B. C. Turner, et al. (1976). “Strains of Neurospora collected from nature.” Evolution 30: 281-313.
Ramos, J., K. Szkutnicka, and V. P. Cirillo (1988). “Relationship between low- and high-affinity glucose transport systems of Saccharomyces cerevisiae.” J Bacteriol 170(11):5375-5377.
Rawat, U. and M. Rao (1997). “Site and significance of cysteine residues in xylose reductase from Neurospora crassa as deduced by fluorescence studies.” Biochemical and Biophysical Research Communications 239(3): 789-93.
Reifenberger E., E. Boles, and M. Ciriacy (1997). “Kinetic characterization of individual hexose transporters of Saccharomyces cerevisiae and their relation to the triggering mechanisms of glucose repression.” Eur J Biochem 245(2):324-333.
Romero, M. D., J. Aguado, et al. (1999). “Cellulase production by Neurospora crassa on wheat straw.” Enzyme Microb Tech 25: 244-250.
Roy, A., A. Kucukural, and Y. Zhang (2010). “I-TASSER: a unified platform for automated protein structure and function prediction.” Nat Protoc 5(4):725-738.
Rubin E. M. (2008). “Genomics of cellulosic biofuels.” Nature 454(7206):841-845.
Ruepp, A., A. Zollner, et al. (2004). “The FunCat, a functional annotation scheme for systematic classification of proteins from whole genomes.” Nucleic Acids Res 32(18):5539-5545.
Runquist, D., C. Fonseca, et al. (2009). “Expression of the Gxf1 transporter from Candida intermedia improves fermentation performance in recombinant xylose-utilizing Saccharomyces cerevisiae.” Appl Microbiol Biotechnol 82(1):123-130.
Runquist, D., B. Hahn-Hagerdal, and P. Radstrom (2010). “Comparison of heterologous xylose transporters in recombinant Saccharomyces cerevisiae.” Biotechnol Biofuels 3:5.
Saha, B. C. (2003). “Hemicellulose bioconversion.” J IndMicrobiol Biotechnol 30:279-291.
Saloheimo, A., J. Rauta, et al. (2007). “Xylose transport studies with xylose-utilizing Saccharomyces cerevisiae strains expressing heterologous and homologous permeases.” Appl Microbiol Biotechnol 74(5):1041-1052.
Sarkar, G. and S. S. Sommer (1990). “The “megaprimer" method of site-directed mutagenesis.” Biotechniques 8(4):404-407.
Sauer, U. (2001). “Evolutionary engineering of industrially important microbial phenotypes.” Adv Biochem Eng Biotechnol 73:129-169.
Scarborough, G. A. (1973). “Transport in Neurospora.” Int Rev Cytol 34:103-122.
Seiboth B, S. Hakola, et al. (1997). “Role of four major cellulases in triggering of cellulase gene expression by cellulose in Trichoderma reesei.” J Bacteriol 179(17):5318-5320.
Seidl, V., C. Gamauf, et al. (2008). “The Hypocrea jecorina (Trichoderma reesei) hypercellulolytic mutant RUT C30 lacks a 85 kb (29 gene-encoding) region of the wild-type genome.” BMC Genomics 9:327.
Shao, Z., H. Zhao, and H. Zhao (2009). “DNA assembler, an in vivo genetic method for rapid construction of biochemical pathways.” Nucleic Acids Res. 37(2):e16.
Sikorski, R. S. and P. Hieter (1989). “A system of shuttle vectors and yeast host strains designed for efficient manipulation of DNA in Saccharomyces cerevisiae.” Genetics 122(1):19-27.
Skory, C. D., S. N. Freer, and R. J. Bothast (1996). “Expression and secretion of the Candida wickerhamii extracellular beta-glucosidase gene, bglB, in Saccharomyces cerevisiae.” Curr Genet 30(5):417-422.
Smith, M. L., O. C. Micali, et al. (2000). “Vegetative incompatibility in the het-6 region of Neurospora crassa is mediated by two linked genes.” Genetics 155(3):1095-1104.
Stambuk, B. U., M. A. Franden, et al. (2003). “D-Xylose transport by Candida succiphila and Kluyveromyces marxianus.” Appl Biochem Biotechnol 105-108:255-263.
Stephanopoulos, G. (2007). “Challenges in Engineering Microbes for Biofuels Production.” Science 315(5813):801-804.
Stricker A.R., R. L. Mach and L. H. de Graaff (2008). “Regulation of transcription of cellulases- and hemicellulases-encoding genes in Aspergillus niger and Hypocrea jecorina (Trichoderma reesei).” Appl Microbiol Biotechnol 78(2):211-220.
Sullivan, R. and H. Zhao (2007). “Cloning, characterization, and mutational analysis of a highly active and stable L-arabinitol 4-dehydrogenase from Neurospora crassa.” Appl Microbiol Biotechnol 77(4):845-52.
Sun Y. and J. Cheng (2002). “Hydrolysis of lignocellulosic materials for ethanol production: a review.” Bioresource Technol 83(1):1-11.
Suominen P. L., A. L. Mantyla, et al. (1993). “High frequency one-step gene replacement in Trichoderma reesei. II. Effects of deletions of individual cellulase genes.” Mol Gen Genet 241(5-6):523-530.
Tian, C., W. T. Beeson, et al. (2009). “Systems analysis of plant cell wall degradation by the model filamentous fungus Neurospora crassa.” Proc Natl Acad Sci USA 106(52):22157-22162.
Tian, C., T. Kasuga, et al. (2007). “Transcriptional profiling of cross pathway control in Neurospora crassa and comparative analysis of the Gcn4 and CPC1 regulons.” Eukaryot Cell 6(6): 1018-1029.
Townsend J. P. (2004). “Resolution of large and small differences in gene expression using models for the Bayesian analysis of gene expression levels and spotted DNA microarrays.” BMC Bioinformatics 5:54.
Townsend J. P. and D. L. Hartl (2002). “Bayesian analysis of gene expression levels: statistical quantification of relative mRNA level across multiple strains or treatments.” Genome Biol 3(12):research0071.1 -0071.16.
USDA, The Economic Feasibility of Ethanol Production from Sugar in the United States, United States Department of Agriculture (2006).
van Rooyen, R., B. Hahn-Hagerdal, et al. (2005). “Construction of cellobiose-growing and fermenting Saccharomyces cerevisiae strains.” J Biotechnol 120(3):284-295.
Vogel, H. J. (1956). “A convenient growth medium for Neurospora.” Microbiol. Genet. Bull. 13:42-46.
Wagner, S., M. L. Bader, et al. (2006). “Rationalizing membrane protein overexpression.” Trends Biotechnol 24(8):364-371.
Watanabe, S., A. Abu Saleh, et al. (2007). “Ethanol production from xylose by recombinant Saccharomyces cerevisiae expressing protein-engineered NADH-preferring xylose reductase from Pichia stipitis.” Microbiology 153(Pt 9):3044-3054.
Watanabe, S., T. Kodaki, and K. Makino (2005a). “L-Arabinose 1-dehydrogenase: a novel enzyme involving in bacterial L-arabinose metabolism.” Nucleic Acids Symp Ser (Oxf) 49:309-310.
Watanabe, S., T. Kodaki, and K. Makino (2005b). “Complete reversal of coenzyme specificity of xylitol dehydrogenase and increase of thermostability by the introduction of structural zinc.” J Biol Chem 280(11):10340-10349.
Wiedemann, B. and E. Boles (2008). “Codon-optimized bacterial genes improve L-arabinose fermentation in recombinant Saccharomyces cerevisiae.” Appl Environ Microbiol 74(7):2043-2050.
Wisselink, H. W., M. J. Toirkens, et al. (2007). “Engineering of Saccharomyces cerevisiae for efficient anaerobic alcoholic fermentation of L-arabinose.” Appl Environ Microbiol 73(15):4881-4891.
Wisselink, H. W., M. J. Toirkens, et al. (2009). “Novel Evolutionary Engineering Approach for Accelerated Utilization of Glucose, Xylose, and Arabinose Mixtures by Engineered Saccharomyces cerevisiae Strains.” Appl Environ Microbiol 75(4):907-914.
Woodyer, R., M. Simurdiak, et al. (2005). “Heterologous expression, purification, and characterization of a highly active xylose reductase from Neurospora crassa.” Appl Environ Microbiol 71(3): 1642-7.
Wymelenberg, A.V., et al. (2009). “Transcriptome and Secretome Analyses of Phanerochaete Chrysosporium Reveal Complex Patterns of Gene Expression.” Appl Environ Microbiol 75(12):4058-4068.
Xin, Z, Q. Yinbo, and G. Peiji. (1993). “Acceleration of ethanol production from paper mill waste fiber by supplementation with β-glucosidase.” Enzyme Microb Tech 15(1):62.
Arendt, C. S., K. Ri, et al. (2007). “Genetic selection for a highly functional cysteine-less membrane protein using site saturation mutagenesis.” Anal Biochem 365(2):185-193.
Bai, F. W., W. A. Anderson, and M. Moo-Young (2008). “Ethanol fermentation technologies from sugar and starch feedstocks.” Biotechnol Adv 26(1): 89-105.
Bailey, M. J., P. Biely, and K. Poutanen (1992). “Interlaboratory testing of methods for assay of xylanase activity.” J Biotechnol 23:257-270.
Bailey, J. E. and D. F. Ollis (1986). Biochemical Engineering Fundamentals, McGraw-Hill Book Company, NY.
Basso, L. C., H. V. de Amorim, et al. (2008). “Yeast selection for fuel ethanol production in Brazil.” FEMS Yeast Res 8(7):1155-1163.
Becker, D. M. and V. Lundblad (2001). “Introduction of DNA into yeast cells.” Curr Protoc Mol Biol Chapter 13, Unit13.7.
Bendtsen, J. D., H. Nielsen, et al. (2004). “Improved prediction of signal peptides: SignalP 3.0.” J. Mol. Biol 340(4):783-95.
Bengsston, O., B. Hahn-Hagerdal, and M. F. Gorwa-Grauslund (2009). “Xylose reductase from Pichia stipitis with altered coenzyme preference improves ethanolic xylose fermentation by recombinant Saccharomyces cerevisiae.” Biotechnol Biofuels 2:9.
Bhat, K. M. and R. Maheshwari (1987). “Sporotrichum thermophile Growth, Cellulose Degradation, and Cellulase Activity.” Appl Environ Microbiol 53(9):2175-2182.
Bouffard, G. G., K. E. Rudd, and S. L. Adhya (1994). “Dependence of lactose metabolism upon mutarotase encoded in the gal operon in Escherichia coli.” J Mol Biol 244(3):269-278.
Bouws, H, A. Wattenberg and H. Zorn (2008). “Fungal secretomes--nature's toolbox for white biotechnology.” Appl Microbiol Biotechnol 80(3):381-388.
Brat, D., E. Boles, and B. Wiedemann (2009). “Functional Expression of a Bacterial Xylose Isomerase in Saccharomyces cerevisiae.” Appl Environ Microbiol 75(8):2304-2311.
Canevascini, G. (1988). “Cellobiose dehydrogenase from Sporotrichum thermophile.” Method Enzymol 160:443-448.
Cantarel, B. L., P. M. Coutinho, et al. (2009). “The Carbohydrate-Active EnZymes database (CAZy): an expert resource for glycogenomics.” Nucleic Acids Res 37(Database issue):D233-238.
Chauve, M., H. Mathis, et al. (2010). “Comparative kinetic analysis of two fungal beta-glucosidases.” Biotechnol Biofuels 3(1):3.
Christakopoulos, P., M. K. Bhat, et al. (1994). “Enzymatic synthesis of trisaccharides and alkyl beta-D-glucosides by the transglycosylation reaction of beta-glucosidase from Fusarium oxysporum.” Int J Biol Macromol 16(6):331-334.
Christianson, T.W., R. S. Sikorskim, et al., (1992). “Multifunctional yeast high-copy number shuttle vectors.” Gene, 110(1):119-122.
Chu, B. C. and H. Lee (2007). “Genetic improvement of Saccharomyces cerevisiae for xylose fermentation.” Biotechnol Adv 25(5):425-441.
Colot, H. V., G. Park, et al. (2006). “A high-throughput gene knockout procedure for Neurospora reveals functions for multiple transcription factors.” Proc Natl Acad Sci USA 103(27):10352-10357.
Cullen, D., L. J. Wilson, et al. (1987). “Sequence and centromere proximal location of a transformation enhancing fragment ans1 from Aspergillus nidulans.” Nucleic Acids Res 15(22):9163-9175.
Davies, G. J., A. M. Brzozowski, et al. (2000).”Structure and function of Humicola insolens family 6 cellulases: structure of the endoglucanase, Cel6B, at 1.6 A resolution.” Biochem J 348 Pt 1:201-207.
Davis, R. H. (2000). “Neurospora: Contributions of a model organism.” New York, Oxford UniversityPress.
Davis, R. H. and D. D. Perkins (2002). “Timeline: Neurospora: a model of model microbes.” Nat Rev Genet 3(5):397-403.
de Groot, M. J., W. Prathumpai, et al. (2005). “Metabolic control analysis of Aspergillus niger L-arabinose catabolism.” Biotechnol Prog 21(6):1610-1616.
Dementhon K, G. Iyer, and N. L. Glass (2006). “VIB-1 is required for expression of genes necessary for programmed cell death in Neurospora crassa.” Eukaryot Cell 5(12):2161-2173.
Doran-Peterson, J., A. Jangid, et al. (2009). “Simultaneous saccharification and fermentation and partial saccharification and co-fermentation of lignocellulosic biomass for ethanol production.” Methods Mol Biol 581:263-280.
Drissen, R. E. T., R. H. W. Mass, et al. (2009). “Modelling ethanol production from cellulose: separate hydrolysis and fermentation versus simultaneous saccharification and fermentation.” Biocatal Biotransfor 27(1):27-35.
Dunlap, J. C., K. A. Borkovich, et al. (2007). “Enabling a Community to Dissect an Organism: Overview of the Neurospora Functional Genomics Project.” Adv Genet 57:49-96.
du Preez, J. C., M. Bosch, and B. A. Prior (1986). “The fermentation of hexose and
pentose sugars by Candida shehatae and Pichia stipitis.” Appl Microbiol Biotechnol 23(3):228-233.
Eberhart, B. M., R. S. Beck, et al. (1977). “Cellulase of Neurospora crassa.” J Bacteriol 130(1):181-186.
Ellis, K. J. and J. F. Morrison (1982). “Buffers of constant ionic strength for studying pH-dependent processes.” Methods Enzymol 87:405-426.
Espagne, E., O. Lespinet, et al. (2008). “The genome sequence of the model ascomycete fungus Podospora anserina.” Genome Biol 9(5):R77.
Fonseca, C. R. Romao, et al. (2007). “L-Arabinose transport and catabolism in yeast.” FEBS J 274(14):3589-3600.
Freer, S. N. (1991). “Fermentation and aerobic metabolism of cellodextrins by yeasts.” Appl Environ Microbiol 57(3):655-659.
Freer, S. N. and R. V. Greene (1990). “Transport of glucose and cellobiose by Candida wickerhamii and Clavispora lusitaniae.” J Biol Chem 265(22):12864-12868.
Galagan, J. E., S. E. Calvo, et al. (2003). “The genome sequence of the filamentous fungus Neurospora crassa.” Nature 422(6934):859-868.
Gems, D., I.L. Johnstone, and A. J. Clutterbuck (1991). “An autonomously replicating plasmid transforms Aspergillus nidulans at high frequency.” Gene 98(1):61-67.
Guindon, S. and O. Gascuel (2003). “A simple, fast, and accurate algorithm to estimate large phylogenies by maximum likelihood.” Syst Biol 52(5):696-704.
Hahn-Hagerdal, B., K. Karhumaa, et al. (2007). “Towards industrial pentose-fermenting yeast strains.” Appl Microbiol Biotechnol 74(5):937-953.
Hector, R. E., N. Qureshi et al. (2008). “Expression of a heterologous xylose transporter in a Saccharomyces cerevisiae strain engineered to utilize xylose improves aerobic xylose consumption.” Appl Microbiol Biotechnol 80(4):675-684.
Himmel, M. E., S. Y. Ding, et al. (2007). “Biomass recalcitrance: engineering plants and enzymes for biofuels production.” Science 315(5813):804-807.
Hosaka, K., J. Nikawa, et al., (1992). “A dominant mutation that alters the regulation of INO1 expression in Saccharomyces cerevisiae.” J Biochem 111(3):352-358.
Jacobson, D. J., A. J. Powell, et al. (2004). “Neurospora in temperate forests of western North America.” Mycologia 96(1):66-74.
Jeffries, T. W., I.V. Grigoriev, et al. (2007). “Genome sequence of the lignocellulose-bioconverting and xylose-fermenting yeast Pichia stipitis.” Nat Biotechnol 25(3):319-326.
Kasuga, T. and N. L. Glass (2008). “Dissecting colony development of Neurospora crassa using mRNA profiling and comparative genomics approaches.” Eukaryot Cell 7(9): 1549-64.
Kasuga, T., J. P. Townsend, et al. (2005). “Long-oligomer microarray profiling in Neurospora crassa reveals the transcriptional program underlying biochemical and physiological events of conidial germination.” Nucleic Acids Res 33(20): 6469-85.
Katahira, S., M. Ito, et al. (2008). “Improvement of ethanol productivity during xylose and glucose co-fermentation by xylose-assimilating S. cerevisiae via expression of glucose transporter Sut1.” Enzyme Microb Tech 43(2):115-119.
Katahira, S., A. Mizuike, et al. (2006). “Ethanol fermentation from lignocellulosic hydrolysates by a recombinant xylose- and cellooligosaccharide-assimilating yeast strain.” Appl Microbiol Biotechnol 72(6):1136-1143.
Korkhin, Y., A. J. Kalb(Gilboa), et al. (1998). “NADP-dependent bacterial alcohol dehydrogenases: crystal structure, cofactor-binding and cofactor specificity of the ADHs of Clostridium beijerinckii and Thermoanaerobacter brockii.” J Mol Biol 278(5):967-981.
Kotaka, A., H. Bando, et al. (2008). “Direct ethanol production from barley beta-glucan by sake yeast displaying Aspergillus oryzae beta-glucosidase and endoglucanases.” J Biosci Bioeng 105(6):622-627.
Kotter, P. and M. Ciriacy (1993). “Xylose fermentation by Saccharomyces cerevisiae.” Appl Microbiol Biotechnol 38(6):776-783.
Kubicek, C. P., R. Messner, et al. (1993). “Triggering of cellulase biosynthesis by cellulose in Trichoderma reesei. Involvement of a constitutive, sophorose-inducible, glucose-inhibited beta-diglucoside permease.” J Biol Chem 268(26):19364-19368.
Kumar, R., S. Singh, and O. V. Singh (2008). “Bioconversion of lignocellulosic biomass: biochemical and molecular perspectives.” J IndMicrobiol Biotechnol 35(5):377-391.
Kuyper, M, H. R. Harhangi, et al. (2003). “High-level functional expression of a fungal xylose isomerase: the key to efficient ethanolic fermentation of xylose by Saccharomyces cerevisiae? FEMS Yeast Res 4(1):69-78.
Lang, J. M. and V. P. Cirillo (1987). “Glucose transport in a kinaseless Saccharomyces cerevisiae mutant.” J Bacteriol 169(7):2932-2937.
Leandro, M. J., P. Gonsalves, and I. Spencer-Martins (2006). “Two glucose/xylose transporter genes from the yeast Candida intermedia: first molecular characterization of a yeast xylose-H+ symporter.” Biochem J 395(3):543-549.
Linder, M. and T. T. Teeri (1996). “The cellulose-binding domain of the major cellobiohydrolase of Trichoderma reesei exhibits true reversibility and a high exchange rate on crystalline cellulose.” Proc Natl Acad Sci USA93(22):12251-12255.
Lynd, L. R., M. S. Laser, et al. (2008). “How biotech can transform biofuels.” Nat. Biotechnol. 26(2):169-172.
Lynd, L. R., P. J. Weimer, et al. (2002). “Microbial cellulose utilization: fundamentals and biotechnology.” Microbiol Mol Biol Rev 66(3):506-77, table of contents.
Madhavan, A., S. Tamalampudi, et al. (2009). “Xylose isomerase from polycentric fungus orpinomyces: gene sequencing, cloning, and expression in Saccharomyces cerevisiae for bioconversion of xylose to ethanol.” Appl Microbiol Biotechnol 82(6):1067-1078.
Martin, F., A. Kohler, et al. (2010). “Perigord black truffle genome uncovers evolutionary origins and mechanisms of symbiosis.” Nature 464(7291):1033-1038.
Martinez, D., R. M. Berka, et al. (2008). “Genome sequencing and analysis of the biomass-degrading fungus Trichoderma reesei (syn. Hypocrea jecorina).” Nat Biotechnol 26(5):553-60.
Martinez, D., J. Callacombe, et al. (2009). “Genome, transcriptome, and secretome analysis of wood decay fungus Postia placenta supports unique mechanisms of lignocellulose conversion.” Proc Natl Acad Sci USA106(6):1954-1959.
Martinez, D., L. F. Larrondo, et al. (2004). “Genome sequence of the lignocellulose degrading fungus Phanerochaete chrysosporium strain RP78.” Nat Biotechnol 22(6):695-700.
Matsushika, A., S. Watanabe, et al. (2008). “Expression of protein engineered NADP+-dependent xylitol dehydrogenase increases ethanol production from xylose in recombinant Saccharomyces cerevisiae.” Appl Microbiol Biotechnol 81(2):243-255.
Matteucci, M. D. and M. H. Caruthers (1980). Tetrahedron Lett 21:719-722.
McCluskey, K. (2003). “The Fungal Genetics Stock Center: from molds to molecules.” Adv Appl Microbiol 52:245-262.
Medve, J., J. Karlsson, et al. (1998). “Hydrolysis of microcrystalline cellulose by cellobiohydrolase I and endoglucanase II from Trichoderma reesei: adsorption, sugar production pattern, and synergism of the enzymes.” Biotechnol Bioeng 59(5):621-34.
Miyasaka, H. (1999). “The positive relationship between codon usage bias and translation initiation AUG context in Saccharomyces cerevisiae.” Yeast 15(8):633-637.
Nair, N. and H. Zhao (2007). “Biochemical characterization of an L-Xylulose reductase from Neurospora crassa.” Appl Environ Microbiol 73(6):2001-2004.
Nakamura, N., R. Yamada, et al. (2008). “Effective xylose/cellobiose co-fermentation and ethanol production by xylose-assimilating S. cerevisiae via expression of β-glucosidase on its cell surface.” Enzyme Microb Tech 43(3):233-236.
Noguchi, Y., M. Sano, et al. (2009). “Genes regulated by AoXlnR, the xylanolytic and cellulolytic transcriptional regulator, in Aspergillus oryzae.” Appl Microbiol Biotechnol 85(1):141-154.
Notredame, C., D. G. Higgins, and J. Heringa (2000). “T-Coffee: A novel method for fast and accurate multiple sequence alignment.” J Mol Biol 302(1):205-217.
Pandit, A. and R. Maheshwari (1996). “Life-history of Neurospora intermedia in a sugar cane field.” J Biosci (Bangalore) 21(1):57-79.
Pauly, T. A., J. L. Ekstrom, et al. (2003). “X-ray crystallographic and kinetic studies of human sorbitol dehydrogenase.” Structure 11(9):1071-1085.
Pedelacq, J. D., S. Cabantous, et al. (2006). “Engineering and characterization of a superfolder green fluorescent protein.” Nat Biotechnol 24(1):79-88.
Perkins, D. D., B. C. Turner, et al. (1976). “Strains of Neurospora collected from nature.” Evolution 30: 281-313.
Ramos, J., K. Szkutnicka, and V. P. Cirillo (1988). “Relationship between low- and high-affinity glucose transport systems of Saccharomyces cerevisiae.” J Bacteriol 170(11):5375-5377.
Rawat, U. and M. Rao (1997). “Site and significance of cysteine residues in xylose reductase from Neurospora crassa as deduced by fluorescence studies.” Biochemical and Biophysical Research Communications 239(3): 789-93.
Reifenberger E., E. Boles, and M. Ciriacy (1997). “Kinetic characterization of individual hexose transporters of Saccharomyces cerevisiae and their relation to the triggering mechanisms of glucose repression.” Eur J Biochem 245(2):324-333.
Romero, M. D., J. Aguado, et al. (1999). “Cellulase production by Neurospora crassa on wheat straw.” Enzyme Microb Tech 25: 244-250.
Roy, A., A. Kucukural, and Y. Zhang (2010). “I-TASSER: a unified platform for automated protein structure and function prediction.” Nat Protoc 5(4):725-738.
Rubin E. M. (2008). “Genomics of cellulosic biofuels.” Nature 454(7206):841-845.
Ruepp, A., A. Zollner, et al. (2004). “The FunCat, a functional annotation scheme for systematic classification of proteins from whole genomes.” Nucleic Acids Res 32(18):5539-5545.
Runquist, D., C. Fonseca, et al. (2009). “Expression of the Gxf1 transporter from Candida intermedia improves fermentation performance in recombinant xylose-utilizing Saccharomyces cerevisiae.” Appl Microbiol Biotechnol 82(1):123-130.
Runquist, D., B. Hahn-Hagerdal, and P. Radstrom (2010). “Comparison of heterologous xylose transporters in recombinant Saccharomyces cerevisiae.” Biotechnol Biofuels 3:5.
Saha, B. C. (2003). “Hemicellulose bioconversion.” J IndMicrobiol Biotechnol 30:279-291.
Saloheimo, A., J. Rauta, et al. (2007). “Xylose transport studies with xylose-utilizing Saccharomyces cerevisiae strains expressing heterologous and homologous permeases.” Appl Microbiol Biotechnol 74(5):1041-1052.
Sarkar, G. and S. S. Sommer (1990). “The “megaprimer" method of site-directed mutagenesis.” Biotechniques 8(4):404-407.
Sauer, U. (2001). “Evolutionary engineering of industrially important microbial phenotypes.” Adv Biochem Eng Biotechnol 73:129-169.
Scarborough, G. A. (1973). “Transport in Neurospora.” Int Rev Cytol 34:103-122.
Seiboth B, S. Hakola, et al. (1997). “Role of four major cellulases in triggering of cellulase gene expression by cellulose in Trichoderma reesei.” J Bacteriol 179(17):5318-5320.
Seidl, V., C. Gamauf, et al. (2008). “The Hypocrea jecorina (Trichoderma reesei) hypercellulolytic mutant RUT C30 lacks a 85 kb (29 gene-encoding) region of the wild-type genome.” BMC Genomics 9:327.
Shao, Z., H. Zhao, and H. Zhao (2009). “DNA assembler, an in vivo genetic method for rapid construction of biochemical pathways.” Nucleic Acids Res. 37(2):e16.
Sikorski, R. S. and P. Hieter (1989). “A system of shuttle vectors and yeast host strains designed for efficient manipulation of DNA in Saccharomyces cerevisiae.” Genetics 122(1):19-27.
Skory, C. D., S. N. Freer, and R. J. Bothast (1996). “Expression and secretion of the Candida wickerhamii extracellular beta-glucosidase gene, bglB, in Saccharomyces cerevisiae.” Curr Genet 30(5):417-422.
Smith, M. L., O. C. Micali, et al. (2000). “Vegetative incompatibility in the het-6 region of Neurospora crassa is mediated by two linked genes.” Genetics 155(3):1095-1104.
Stambuk, B. U., M. A. Franden, et al. (2003). “D-Xylose transport by Candida succiphila and Kluyveromyces marxianus.” Appl Biochem Biotechnol 105-108:255-263.
Stephanopoulos, G. (2007). “Challenges in Engineering Microbes for Biofuels Production.” Science 315(5813):801-804.
Stricker A.R., R. L. Mach and L. H. de Graaff (2008). “Regulation of transcription of cellulases- and hemicellulases-encoding genes in Aspergillus niger and Hypocrea jecorina (Trichoderma reesei).” Appl Microbiol Biotechnol 78(2):211-220.
Sullivan, R. and H. Zhao (2007). “Cloning, characterization, and mutational analysis of a highly active and stable L-arabinitol 4-dehydrogenase from Neurospora crassa.” Appl Microbiol Biotechnol 77(4):845-52.
Sun Y. and J. Cheng (2002). “Hydrolysis of lignocellulosic materials for ethanol production: a review.” Bioresource Technol 83(1):1-11.
Suominen P. L., A. L. Mantyla, et al. (1993). “High frequency one-step gene replacement in Trichoderma reesei. II. Effects of deletions of individual cellulase genes.” Mol Gen Genet 241(5-6):523-530.
Tian, C., W. T. Beeson, et al. (2009). “Systems analysis of plant cell wall degradation by the model filamentous fungus Neurospora crassa.” Proc Natl Acad Sci USA 106(52):22157-22162.
Tian, C., T. Kasuga, et al. (2007). “Transcriptional profiling of cross pathway control in Neurospora crassa and comparative analysis of the Gcn4 and CPC1 regulons.” Eukaryot Cell 6(6): 1018-1029.
Townsend J. P. (2004). “Resolution of large and small differences in gene expression using models for the Bayesian analysis of gene expression levels and spotted DNA microarrays.” BMC Bioinformatics 5:54.
Townsend J. P. and D. L. Hartl (2002). “Bayesian analysis of gene expression levels: statistical quantification of relative mRNA level across multiple strains or treatments.” Genome Biol 3(12):research0071.1 -0071.16.
USDA, The Economic Feasibility of Ethanol Production from Sugar in the United States, United States Department of Agriculture (2006).
van Rooyen, R., B. Hahn-Hagerdal, et al. (2005). “Construction of cellobiose-growing and fermenting Saccharomyces cerevisiae strains.” J Biotechnol 120(3):284-295.
Vogel, H. J. (1956). “A convenient growth medium for Neurospora.” Microbiol. Genet. Bull. 13:42-46.
Wagner, S., M. L. Bader, et al. (2006). “Rationalizing membrane protein overexpression.” Trends Biotechnol 24(8):364-371.
Watanabe, S., A. Abu Saleh, et al. (2007). “Ethanol production from xylose by recombinant Saccharomyces cerevisiae expressing protein-engineered NADH-preferring xylose reductase from Pichia stipitis.” Microbiology 153(Pt 9):3044-3054.
Watanabe, S., T. Kodaki, and K. Makino (2005a). “L-Arabinose 1-dehydrogenase: a novel enzyme involving in bacterial L-arabinose metabolism.” Nucleic Acids Symp Ser (Oxf) 49:309-310.
Watanabe, S., T. Kodaki, and K. Makino (2005b). “Complete reversal of coenzyme specificity of xylitol dehydrogenase and increase of thermostability by the introduction of structural zinc.” J Biol Chem 280(11):10340-10349.
Wiedemann, B. and E. Boles (2008). “Codon-optimized bacterial genes improve L-arabinose fermentation in recombinant Saccharomyces cerevisiae.” Appl Environ Microbiol 74(7):2043-2050.
Wisselink, H. W., M. J. Toirkens, et al. (2007). “Engineering of Saccharomyces cerevisiae for efficient anaerobic alcoholic fermentation of L-arabinose.” Appl Environ Microbiol 73(15):4881-4891.
Wisselink, H. W., M. J. Toirkens, et al. (2009). “Novel Evolutionary Engineering Approach for Accelerated Utilization of Glucose, Xylose, and Arabinose Mixtures by Engineered Saccharomyces cerevisiae Strains.” Appl Environ Microbiol 75(4):907-914.
Woodyer, R., M. Simurdiak, et al. (2005). “Heterologous expression, purification, and characterization of a highly active xylose reductase from Neurospora crassa.” Appl Environ Microbiol 71(3): 1642-7.
Wymelenberg, A.V., et al. (2009). “Transcriptome and Secretome Analyses of Phanerochaete Chrysosporium Reveal Complex Patterns of Gene Expression.” Appl Environ Microbiol 75(12):4058-4068.
Xin, Z, Q. Yinbo, and G. Peiji. (1993). “Acceleration of ethanol production from paper mill waste fiber by supplementation with β-glucosidase.” Enzyme Microb Tech 15(1):62.
Claims (119)
- 細胞へのセロデキストリンの輸送を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記組換えポリヌクレオチドが発現されるような培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス1は配列番号1を含んでおり、
上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記宿主細胞へのセロデキストリンの促進された輸送をもたらす、方法。 - 細胞へのセロデキストリンの輸送を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記組換えポリヌクレオチドが発現されるような培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス2は配列番号2を含んでおり、
上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記宿主細胞へのセロデキストリンの促進された輸送をもたらす、方法。 - 細胞へのセロデキストリンの輸送を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記組換えポリヌクレオチドが発現されるような培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス2および膜貫通型のα−ヘリックス3を接続しているループは配列番号3を含んでおり、
上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記宿主細胞へのセロデキストリンの促進された輸送をもたらす、方法。 - 細胞へのセロデキストリンの輸送を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記組換えポリヌクレオチドが発現されるような培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス5は配列番号4を含んでおり、
上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記宿主細胞へのセロデキストリンの促進された輸送をもたらす、方法。 - 細胞へのセロデキストリンの輸送を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記組換えポリヌクレオチドが発現されるような培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス6は配列番号5を含んでおり、
上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記宿主細胞へのセロデキストリンの促進された輸送をもたらす、方法。 - 細胞へのセロデキストリンの輸送を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記組換えポリヌクレオチドが発現されるような培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス6および膜貫通型のα−ヘリックス7の間にある領域は配列番号6を含んでおり、
上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記宿主細胞へのセロデキストリンの促進された輸送をもたらす、方法。 - 細胞へのセロデキストリンの輸送を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記組換えポリヌクレオチドが発現されるような培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス7は配列番号7を含んでおり、
上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記宿主細胞へのセロデキストリンの促進された輸送をもたらす、方法。 - 細胞へのセロデキストリンの輸送を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記組換えポリヌクレオチドが発現されるような培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびこれらの間にある配列は、配列番号8を含んでおり、
上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記宿主細胞へのセロデキストリンの促進された輸送をもたらす、方法。 - 上記ポリペプチドは、少なくとも29%、少なくとも30%、少なくとも35%、少なくとも40%、少なくとも45%、少なくとも50%、少なくとも55%、少なくとも60%、少なくとも65%、少なくとも70%、少なくとも75%、少なくとも80%、少なくとも85%、少なくとも90%、少なくとも95%、少なくとも99%または少なくとも100%の、NCU00801またはNCU08114に対するアミノ酸同一性を有している、請求項1〜8のいずれか1項に記載の方法。
- 上記宿主細胞は、β−グルコシダーゼの触媒作用ドメインを少なくともコードしている第2の組換えポリヌクレオチドをさらに含んでいる、請求項1〜9のいずれか1項に記載の方法。
- 上記β−グルコシダーゼはNeurospora crassaに由来する、請求項10に記載の方法。
- 上記β−グルコシダーゼはNCU00130によってコードされている、請求項11に記載の方法。
- 上記宿主細胞は、ペントース利用に関与する1つ以上の酵素をコードしている1つ以上の組換えポリヌクレオチドをさらに含んでいる、請求項1〜12のいずれか1項に記載の方法。
- 1つ以上の上記酵素は、L−アラビノースイソメラーゼ、L−リブロキナーゼ、L−リブロース−5−P 4 エピメラーゼ、キシロースイソメラーゼ、キシルロキナーゼ、アルドースリダクターゼ、L−アラビニトール 4−デヒドロゲナーゼ、L−キシルロースリダクターゼ、およびキシトールデヒドロゲナーゼからなる群のうちの1つ以上から選択される、請求項13に記載の方法。
- 上記細胞は、ペントーストランスポータをコードしている第3の組換えポリヌクレオチドをさらに含んでいる、請求項1〜14のいずれか1項に記載の方法。
- 上記ペントーストランスポータは、NCU00821、NCU04963、NCU06138、STL12/XUT6、SUT2、SUT3、XUT1、およびXUT3からなる群から選択される、請求項15に記載の方法。
- 細胞の成長を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記組換えポリヌクレオチドが発現されるような培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス1は配列番号1を含んでおり、上記ポリペプチドはセロデキストリントランスポータであり、
上記宿主細胞は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する、方法。 - 細胞の成長を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記組換えポリヌクレオチドが発現されるような培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス2は配列番号2を含んでおり、上記ポリペプチドはセロデキストリントランスポータであり、
上記宿主細胞は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する、方法。 - 細胞の成長を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記組換えポリヌクレオチドが発現されるような培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス2および膜貫通型のα−ヘリックス3を接続しているループは配列番号3を含んでおり、上記ポリペプチドはセロデキストリントランスポータであり、
上記宿主細胞は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する、方法。 - 細胞の成長を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記組換えポリヌクレオチドが発現されるような培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス5は配列番号4を含んでおり、上記ポリペプチドはセロデキストリントランスポータであり、
上記宿主細胞は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する、方法。 - 細胞の成長を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記組換えポリヌクレオチドが発現されるような培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス6は配列番号5を含んでおり、上記ポリペプチドはセロデキストリントランスポータであり、
上記宿主細胞は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する、方法。 - 細胞の成長を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記組換えポリヌクレオチドが発現されるような培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス6および膜貫通型のα−ヘリックス7の間にある領域は配列番号6を含んでおり、上記ポリペプチドはセロデキストリントランスポータであり、
上記宿主細胞は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する、方法。 - 細胞の成長を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記組換えポリヌクレオチドが発現されるような培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス7は配列番号7を含んでおり、上記ポリペプチドはセロデキストリントランスポータであり、
上記宿主細胞は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する、方法。 - 細胞の成長を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記組換えポリヌクレオチドが発現されるような培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびこれらの間にある配列は、配列番号8を含んでおり、上記ポリペプチドはセロデキストリントランスポータであり、
上記宿主細胞は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する、方法。 - 上記ポリペプチドは、少なくとも29%、少なくとも30%、少なくとも35%、少なくとも40%、少なくとも45%、少なくとも50%、少なくとも55%、少なくとも60%、少なくとも65%、少なくとも70%、少なくとも75%、少なくとも80%、少なくとも85%、少なくとも90%、少なくとも95%、少なくとも99%または少なくとも100%の、NCU00801またはNCU08114に対するアミノ酸同一性を有している、請求項17〜24のいずれか1項に記載の方法。
- 上記宿主細胞は、β−グルコシダーゼの触媒作用ドメインを少なくともコードしている外因性または第2の組換えポリヌクレオチドをさらに含んでいる、請求項17〜25のいずれか1項に記載の方法。
- 上記β−グルコシダーゼはNeurospora crassaに由来する、請求項26に記載の方法。
- 上記β−グルコシダーゼはNCU00130によってコードされている、請求項27に記載の方法。
- セルロース由来の糖およびヘミセルロース由来の糖を共発酵させる方法であって、
セロデキストリントランスポータをコードしている第1の組換えポリヌクレオチドおよびβ−グルコシダーゼの触媒作用ドメインをコードしている第2の組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;ならびに
セルロース由来の糖およびヘミセルロース由来の糖を含んでいる培地において上記宿主細胞を培養することを包含しており、
2つの上記組換えポリヌクレオチドの発現がセルロース由来の糖およびヘミセルロース由来の糖の共発酵を可能にする、方法。 - 上記第1の組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス1は配列番号1を含んでいる、請求項29に記載の方法。
- 上記第1の組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス2は配列番号2を含んでいる、請求項29に記載の方法。
- 上記第1の組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス2および膜貫通型のα−ヘリックス3を接続しているループは配列番号3を含んでいる、請求項29に記載の方法。
- 上記第1の組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス5は配列番号4を含んでいる、請求項29に記載の方法。
- 上記第1の組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス6は配列番号5を含んでいる、請求項29に記載の方法。
- 上記第1の組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス6および膜貫通型のα−ヘリックス7の間にある領域は配列番号6を含んでいる、請求項29に記載の方法。
- 上記第1の組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス7は配列番号7を含んでいる、請求項29に記載の方法。
- 上記第1の組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびこれらの間にある配列は、配列番号8を含んでいる、請求項29に記載の方法。
- 上記ポリペプチドは、少なくとも29%、少なくとも30%、少なくとも35%、少なくとも40%、少なくとも45%、少なくとも50%、少なくとも55%、少なくとも60%、少なくとも65%、少なくとも70%、少なくとも75%、少なくとも80%、少なくとも85%、少なくとも90%、少なくとも95%、少なくとも99%または少なくとも100%の、NCU00801またはNCU08114に対するアミノ酸同一性を有している、請求項30〜37のいずれか1項に記載の方法。
- 上記β−グルコシダーゼはNeurospora crassaに由来する、請求項29〜38のいずれか1項に記載の方法。
- 上記β−グルコシダーゼはNCU00130によってコードされている、請求項39に記載の方法。
- 上記宿主細胞は、ペントース利用に関与する1つ以上の酵素をコードしている1つ以上の組換えポリヌクレオチドをさらに含んでいる、請求項29〜40のいずれか1項に記載の方法。
- 1つ以上の上記酵素は、L−アラビノースイソメラーゼ、L−リブロキナーゼ、L−リブロース−5−P 4 エピメラーゼ、キシロースイソメラーゼ、キシルロキナーゼ、アルドースリダクターゼ、L−アラビニトール 4−デヒドロゲナーゼ、L−キシルロースリダクターゼ、およびキシトールデヒドロゲナーゼからなる群のうちの1つ以上から選択される、請求項41に記載の方法。
- 上記宿主細胞は、ペントーストランスポータをコードしている第3の組換えポリヌクレオチドをさらに含んでいる、請求項29〜42のいずれか1項に記載の方法。
- 上記ペントーストランスポータは、NCU00821、NCU04963、NCU06138、STL12/XUT6、SUT2、SUT3、XUT1、およびXUT3からなる群から選択される、請求項43に記載の方法。
- セルロース由来の上記糖は、セロビオース、セロトリオースおよびセロテトラオースからなる群から選択され、ヘミセルロース由来の上記糖はキシロースである、請求項29〜44のいずれか1項に記載の方法。
- 宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、セロデキストリンまたはセロデキストリン源を含んでいる培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス1は配列番号1を含んでおり、上記ポリペプチドは、炭化水素または炭化水素の誘導体の合成のために上記宿主細胞にセロデキストリンを輸送し、
上記宿主細胞へのセロデキストリン輸送は、上記組換えポリヌクレオチドの発現によって促進される、方法。 - 宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、セロデキストリンまたはセロデキストリン源を含んでいる培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス2は配列番号2を含んでおり、上記ポリペプチドは、炭化水素または炭化水素の誘導体の合成のために上記宿主細胞にセロデキストリンを輸送し、
上記宿主細胞へのセロデキストリン輸送は、上記組換えポリヌクレオチドの発現によって促進される、方法。 - 宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、セロデキストリンまたはセロデキストリン源を含んでいる培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス2および膜貫通型のα−ヘリックス3を接続しているループは配列番号3を含んでおり、上記ポリペプチドは、炭化水素または炭化水素の誘導体の合成のために上記宿主細胞にセロデキストリンを輸送し、
上記宿主細胞へのセロデキストリン輸送は、上記組換えポリヌクレオチドの発現によって促進される、方法。 - 宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、セロデキストリンまたはセロデキストリン源を含んでいる培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス5は配列番号4を含んでおり、上記ポリペプチドは、炭化水素または炭化水素の誘導体の合成のために上記宿主細胞にセロデキストリンを輸送し、
上記宿主細胞へのセロデキストリン輸送は、上記組換えポリヌクレオチドの発現によって促進される、方法。 - 宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、セロデキストリンまたはセロデキストリン源を含んでいる培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス6は配列番号5を含んでおり、上記ポリペプチドは、炭化水素または炭化水素の誘導体の合成のために上記宿主細胞にセロデキストリンを輸送し、
上記宿主細胞へのセロデキストリン輸送は、上記組換えポリヌクレオチドの発現によって促進される、方法。 - 宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、セロデキストリンまたはセロデキストリン源を含んでいる培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス6および膜貫通型のα−ヘリックス7の間にある領域は配列番号6を含んでおり、上記ポリペプチドは、炭化水素または炭化水素の誘導体の合成のために上記宿主細胞にセロデキストリンを輸送し、
上記宿主細胞へのセロデキストリン輸送は、上記組換えポリヌクレオチドの発現によって促進される、方法。 - 宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、セロデキストリンまたはセロデキストリン源を含んでいる培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス7は配列番号7を含んでおり、上記ポリペプチドは、炭化水素または炭化水素の誘導体の合成のために上記宿主細胞にセロデキストリンを輸送し、
上記宿主細胞へのセロデキストリン輸送は、上記組換えポリヌクレオチドの発現によって促進される、方法。 - 宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法であって、
組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;および
上記宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、セロデキストリンまたはセロデキストリン源を含んでいる培地において上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドは、膜貫通型のα−ヘリックス1、膜貫通型のα−ヘリックス2、膜貫通型のα−ヘリックス3、膜貫通型のα−ヘリックス4、膜貫通型のα−ヘリックス5、膜貫通型のα−ヘリックス6、膜貫通型のα−ヘリックス7、膜貫通型のα−ヘリックス8、膜貫通型のα−ヘリックス9、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11、膜貫通型のα−ヘリックス12を含んでいるポリペプチドをコードしており、膜貫通型のα−ヘリックス10、膜貫通型のα−ヘリックス11およびこれらの間にある配列は、配列番号8を含んでおり、上記ポリペプチドは、炭化水素または炭化水素の誘導体の合成のために上記宿主細胞にセロデキストリンを輸送し、
上記宿主細胞へのセロデキストリン輸送は、上記組換えポリヌクレオチドの発現によって促進される、方法。 - 上記ポリペプチドは、少なくとも29%、少なくとも30%、少なくとも35%、少なくとも40%、少なくとも45%、少なくとも50%、少なくとも55%、少なくとも60%、少なくとも65%、少なくとも70%、少なくとも75%、少なくとも80%、少なくとも85%、少なくとも90%、少なくとも95%、少なくとも99%または少なくとも100%の、NCU00801またはNCU08114に対するアミノ酸同一性を有している、請求項46〜53のいずれか1項に記載の方法。
- 上記宿主細胞は、β−グルコシダーゼの触媒作用ドメインを少なくともコードしている第2の組換えポリヌクレオチドをさらに含んでいる、請求項46〜54のいずれか1項に記載の方法。
- 上記β−グルコシダーゼはNeurospora crassaに由来する、請求項55に記載の方法。
- 上記β−グルコシダーゼはNCU00130によってコードされている、請求項56に記載の方法。
- 上記セロデキストリン源はセルロースを含んでいる、請求項46〜57のいずれか1項に記載の方法。
- 上記炭化水素または炭化水素の誘導体は燃料として使用され得る、請求項46〜58のいずれか1項に記載の方法。
- 上記炭化水素または炭化水素の誘導体はエタノールを含んでいる、請求項59に記載の方法。
- 上記炭化水素または炭化水素の誘導体はブタノールを含んでいる、請求項59に記載の方法。
- 上記培地は、改変された生物から得られた、セルラーゼ含有の酵素混合物を含んでおり、セルラーゼ含有の上記酵素混合物は、改変されていない生物から得られた、セルラーゼ含有の酵素混合物と比べて、低下したβ−グルコシダーゼ活性を有している、請求項1〜61のいずれか1項に記載の方法。
- 上記宿主細胞は、Saccharomyces sp.、Saccharomyces cerevisiae、Saccharomyces monacensis、Saccharomyces bayanus、Saccharomyces pastorianus、Saccharomyces carlsbergensis、Saccharomyces pombe、Kluyveromyces sp.、Kluyveromyces marxiamus、Kluyveromyces lactis、Kluyveromyces fragilis、Pichia stipitis、Sporotrichum thermophile、Candida shehatae、Candida tropicalis、Neurospora crassa、Zymomonas mobilis、Clostridium sp.、Clostridium phytofermentans、Clostridium thermocellum、Clostridium beijerinckii、Clostridium acetobutylicum、Moorella thermoacetica、Escherichia coli、Klebsiella oxytoca、Thermoanaerobacterium saccharolyticum、およびBacillus subtilisからなる群から選択される、請求項1〜62のいずれか1項に記載の方法。
- 上記セロデキストリンは、セロビオース、セロトリオースおよびセロテトラオースからなる群のうちの1つ以上から選択される、請求項1〜63のいずれか1項に記載の方法。
- 組換えポリヌクレオチドを含んでいる宿主細胞であって、上記組換えポリヌクレオチドは、NCU00821およびSTL12/XUT6からなる群から選択されるポリペプチドをコードしており、当該ポリペプチドは上記宿主細胞にキシロースを輸送する、宿主細胞。
- 組換えポリヌクレオチドを含んでいる宿主細胞であって、上記組換えポリヌクレオチドはXUT1であるポリペプチドをコードしており、上記ポリペプチドは上記宿主細胞にアラビノースを輸送する、宿主細胞。
- 組換えポリヌクレオチドを含んでいる宿主細胞であって、上記組換えポリヌクレオチドはNCU06138であるポリペプチドをコードしており、上記ポリペプチドは上記宿主細胞にアラビノースおよびグルコースを輸送する、宿主細胞。
- 組換えポリヌクレオチドを含んでいる宿主細胞であって、上記組換えポリヌクレオチドは、SUT2、SUT3およびXUT3からなる群から選択されるポリペプチドをコードしており、上記ポリペプチドは上記宿主細胞にキシロースおよびグルコースを輸送する、宿主細胞。
- 組換えポリヌクレオチドを含んでいる宿主細胞であって、上記組換えポリヌクレオチドはNCU04963であるポリペプチドをコードしており、上記ポリペプチドは上記宿主細胞にキシロース、アラビノースおよびグルコースを輸送する、宿主細胞。
- ペントース利用に関与する1つ以上の酵素をコードしている1つ以上の組換えポリヌクレオチドをさらに含んでいる、請求項65〜69に記載の宿主細胞。
- 1つ以上の上記酵素は、L−アラビノースイソメラーゼ、L−リブロキナーゼ、L−リブロース−5−P 4 エピメラーゼ、キシロースイソメラーゼ、キシルロキナーゼ、アルドースリダクターゼ、L−アラビニトール 4−デヒドロゲナーゼ、L−キシルロースリダクターゼ、およびキシトールデヒドロゲナーゼからなる群のうちの1つ以上から選択される、請求項70に記載の宿主細胞。
- Saccharomyces sp.、Saccharomyces cerevisiae、Saccharomyces monacensis、Saccharomyces bayanus、Saccharomyces pastorianus、Saccharomyces carlsbergensis、Saccharomyces pombe、Kluyveromyces sp.、Kluyveromyces marxiamus、Kluyveromyces lactis、Kluyveromyces fragilis、Pichia stipitis、Sporotrichum thermophile、Candida shehatae、Candida tropicalis、Neurospora crassa、Zymomonas mobilis、Clostridium sp.、Clostridium phytofermentans、Clostridium thermocellum、Clostridium beijerinckii、Clostridium acetobutylicum、Moorella thermoacetica、Escherichia coli、Klebsiella oxytoca、Thermoanaerobacterium saccharolyticum、およびBacillus subtilisからなる群から選択される、請求項65〜71に記載の宿主細胞。
- 細胞へのキシロースの輸送を促進させる方法であって、
NCU00821およびSTL12/XUT6からなる群から選択されるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;
上記組換えポリヌクレオチドが発現されるように上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記宿主細胞への促進されたキシロースの輸送をもたらす、方法。 - 細胞へのアラビノースの輸送を促進させる方法であって、
XUT1であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;
上記組換えポリヌクレオチドが発現されるように上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記宿主細胞への促進されたアラビノースの輸送をもたらす、方法。 - 細胞へのアラビノースまたはグルコースの輸送を促進させる方法であって、
NCU06138であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;
上記組換えポリヌクレオチドが発現されるように上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記宿主細胞への促進されたアラビノースまたはグルコースの輸送をもたらす、方法。 - 細胞へのキシロースまたはグルコースの輸送を促進させる方法であって、
NCU04963であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;
上記組換えポリヌクレオチドが発現されるように上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記宿主細胞への促進されたキシロースまたはグルコースの輸送をもたらす、方法。 - 細胞へのキシロース、アラビノースまたはグルコースの輸送を促進させる方法であって、
NCU04963であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;
上記組換えポリヌクレオチドが発現されるように上記宿主細胞を培養することを包含しており、
上記組換えポリヌクレオチドの発現は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記宿主細胞への促進されたキシロース、アラビノースまたはグルコースの輸送をもたらす、方法。 - ペントース利用に関与する1つ以上の酵素をコードしている1つ以上の組換えポリヌクレオチドをさらに含んでいる、請求項73〜77のいずれか1項に記載の方法。
- 1つ以上の上記酵素は、L−アラビノースイソメラーゼ、L−リブロキナーゼ、L−リブロース−5−P 4 エピメラーゼ、キシロースイソメラーゼ、キシルロキナーゼ、アルドースリダクターゼ、L−アラビニトール 4−デヒドロゲナーゼ、L−キシルロースリダクターゼ、およびキシトールデヒドロゲナーゼからなる群のうちの1つ以上から選択される、請求項78に記載の方法。
- 細胞の成長を促進させる方法であって、
キシロースを輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;ならびに
キシロースを含んでいる培地において上記宿主細胞を培養することを包含しており、
上記ポリペプチドは、NCU00821およびSTL12/XUT6からなる群から選択され、
上記宿主細胞は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する、方法。 - 細胞の成長を促進させる方法であって、
アラビノースを輸送するXUT1であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;ならびに
アラビノースを含んでいる培地において上記宿主細胞を培養することを包含しており、
上記宿主細胞は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する、方法。 - 細胞の成長を促進させる方法であって、
アラビノースおよびグルコースを輸送するNCU06138であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;ならびに
アラビノースまたはグルコースを含んでいる培地において上記宿主細胞を培養することを包含しており、
上記宿主細胞は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する、方法。 - 細胞の成長を促進させる方法であって、
キシロースおよびグルコースを輸送するポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;ならびに
キシロースまたはグルコースを含んでいる培地において上記宿主細胞を培養することを包含しており、
上記ポリペプチドは、SUT2、SUT3およびXUT3からなる群から選択され、
上記宿主細胞は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する、方法。 - 細胞の成長を促進させる方法であって、
キシロース、アラビノースおよびグルコースを輸送するNCU04963であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;ならびに
キシロース、アラビノースまたはグルコースを含んでいる培地において上記宿主細胞を培養することを包含しており、
上記宿主細胞は、当該組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する、方法。 - 上記宿主細胞は、ペントース利用に関与する1つ以上の酵素をコードしている1つ以上の組換えポリヌクレオチドをさらに含んでいる、請求項80〜84のいずれか1項に記載の方法。
- 1つ以上の上記酵素は、L−アラビノースイソメラーゼ、L−リブロキナーゼ、L−リブロース−5−P 4 エピメラーゼ、キシロースイソメラーゼ、キシルロキナーゼ、アルドースリダクターゼ、L−アラビニトール 4−デヒドロゲナーゼ、L−キシルロースリダクターゼ、およびキシトールデヒドロゲナーゼからなる群のうちの1つ以上から選択される、請求項85に記載の方法。
- 宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法であって、
NCU00821およびSTL12/XUT6からなる群から選択されるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;ならびに
上記宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、キシロースまたはキシロース源を含んでいる培地において上記宿主を培養することを包含しており、
上記ポリペプチドは、炭化水素または炭化水素の誘導体の合成のために宿主細胞にキシロースを輸送し、
上記宿主細胞へのキシロースの輸送は、上記組換えポリヌクレオチドの発現によって促進される、方法。 - 宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法であって、
XUT1であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;ならびに
上記宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、アラビノースまたはアラビノース源を含んでいる培地において上記宿主を培養することを包含しており、
上記ポリペプチドは、炭化水素または炭化水素の誘導体の合成のために宿主細胞にアラビノースを輸送し、
上記宿主細胞へのアラビノースの輸送は、上記組換えポリヌクレオチドの発現によって促進される、方法。 - 宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法であって、
NCU06138であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;ならびに
上記宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、アラビノースもしくはキシロースの源、アラビノースまたはグルコースを含んでいる培地において上記宿主を培養することを包含しており、
上記ポリペプチドは、炭化水素または炭化水素の誘導体の合成のために宿主細胞にキシロースまたはグルコースを輸送し、
上記宿主細胞へのアラビノースまたはグルコースの輸送は、上記組換えポリヌクレオチドの発現によって促進される、方法。 - 宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法であって、
SUT2、SUT3およびXUT3からなる群から選択されるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;ならびに
上記宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、キシロースもしくはグルコースの源、キシロースまたはグルコースを含んでいる培地において上記宿主を培養することを包含しており、
上記ポリペプチドは、炭化水素または炭化水素の誘導体の合成のために宿主細胞にキシロースまたはグルコースを輸送し、
上記宿主細胞へのキシロースまたはグルコースの輸送は、上記組換えポリヌクレオチドの発現によって促進される、方法。 - 宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させる方法であって、
NCUポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;ならびに
上記宿主細胞による炭化水素または炭化水素の誘導体の合成を促進させるために、キシロース、アラビノースもしくはグルコースの源、キシロース、アラビノースまたはグルコースを含んでいる培地において上記宿主を培養することを包含しており、
上記ポリペプチドは、炭化水素または炭化水素の誘導体の合成のために宿主細胞にキシロース、アラビノースまたはグルコースを輸送し、
上記宿主細胞へのキシロース、アラビノースまたはグルコースの輸送は、上記組換えポリヌクレオチドの発現によって促進される、方法。 - グルコースの上記源はセルロースを含んでいる、請求項89〜91のいずれか1項に記載の方法。
- キシロースまたはアラビノースの上記源はヘミセルロースを含んでいる、請求項87〜92のいずれか1項に記載の方法。
- 上記炭化水素または炭化水素の誘導体は燃料として使用され得る、請求項87〜93のいずれか1項に記載の方法。
- 上記炭化水素または炭化水素の誘導体はエタノールを含んでいる、請求項94に記載の方法。
- 上記炭化水素または炭化水素の誘導体はブタノールを含んでいる、請求項81に記載の方法。
- 上記宿主細胞は、Saccharomyces sp.、Saccharomyces cerevisiae、Saccharomyces monacensis、Saccharomyces bayanus、Saccharomyces pastorianus、Saccharomyces carlsbergensis、Saccharomyces pombe、Kluyveromyces sp.、Kluyveromyces marxiamus、Kluyveromyces lactis、Kluyveromyces fragilis、Pichia stipitis、Sporotrichum thermophile、Candida shehatae、Candida tropicalis、Neurospora crassa、Zymomonas mobilis、Clostridium sp.、Clostridium phytofermentans、Clostridium thermocellum、Clostridium beijerinckii、Clostridium acetobutylicum、Moorella thermoacetica、Escherichia coli、Klebsiella oxytoca、Thermoanaerobacterium saccharolyticum、およびBacillus subtilisからなる群から選択される、請求項73〜95のいずれか1項に記載の方法。
- 細胞の成長を促進させる方法であって、
NCU07705であるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる細胞を準備すること;および
グルコースを含んでいる培地において上記宿主細胞を培養することを包含しており、
上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、上記培地においてより早く成長する、方法。 - 上記宿主細胞は、Saccharomyces sp.、Saccharomyces cerevisiae、Saccharomyces monacensis、Saccharomyces bayanus、Saccharomyces pastorianus、Saccharomyces carlsbergensis、Saccharomyces pombe、Kluyveromyces sp.、Kluyveromyces marxiamus、Kluyveromyces lactis、Kluyveromyces fragilis、Pichia stipitis、Sporotrichum thermophile、Candida shehatae、Candida tropicalis、Neurospora crassa、Zymomonas mobilis、Clostridium sp.、Clostridium phytofermentans、Clostridium thermocellum、Clostridium beijerinckii、Clostridium acetobutylicum、Moorella thermoacetica、Escherichia coli、Klebsiella oxytoca、Thermoanaerobacterium saccharolyticum、およびBacillus subtilisからなる群から選択される、請求項98に記載の方法。
- 上記宿主細胞は、上記組換えポリヌクレオチドと作動可能に連結されている誘導性のプロモータをさらに含んでいる、請求項98に記載の方法。
- セルラーゼの発現は、上記組換えポリヌクレオチドの発現によって上記宿主細胞において促進されている、請求項98に記載の方法。
- バイオマス重合体に基づく細胞の成長を促進させる方法であって、
NCU05173であるポリペプチドをコードしている外因性のポリヌクレオチドを含んでいる宿主細胞を準備すること;
上記外因性のポリヌクレオチドの発現を抑制すること;および
上記バイオマス重合体を含んでいる培地において上記宿主細胞を培養することを包含しており、
上記宿主細胞は、上記外因性のポリヌクレオチドの発現が抑制されていない細胞と比べて、上記培地においてより早く成長する、方法。 - 上記宿主細胞は、Saccharomyces sp.、Saccharomyces cerevisiae、Saccharomyces monacensis、Saccharomyces bayanus、Saccharomyces pastorianus、Saccharomyces carlsbergensis、Saccharomyces pombe、Kluyveromyces sp.、Kluyveromyces marxiamus、Kluyveromyces lactis、Kluyveromyces fragilis、Pichia stipitis、Sporotrichum thermophile、Candida shehatae、Candida tropicalis、Neurospora crassa、Zymomonas mobilis、Clostridium sp.、Clostridium phytofermentans、Clostridium thermocellum、Clostridium beijerinckii、Clostridium acetobutylicum、Moorella thermoacetica、Escherichia coli、Klebsiella oxytoca、Thermoanaerobacterium saccharolyticum、およびBacillus subtilisからなる群から選択される、請求項102に記載の方法。
- 上記宿主細胞のセルラーゼ活性は上記外因性のポリヌクレオチドの発現を抑制することによって促進される、請求項102に記載の方法。
- 上記宿主細胞のヘミセルラーゼ活性は上記外因性のポリヌクレオチドの発現を抑制することによって促進される、請求項102に記載の方法。
- 上記外因性のポリヌクレオチドの発現を抑制することは、外因性の当該ポリヌクレオチドを含んでいる遺伝子を変異させること、または当該遺伝子を欠失させることを包含している、請求項102に記載の方法。
- 上記バイオマス重合体はセルロースである、請求項102に記載の方法。
- 上記バイオマス重合体はヘミセルロースである、請求項102に記載の方法。
- 細胞の成長を促進させる方法であって、
NCU01517、NCU09133およびNCU10040からなる群から選択されるポリペプチドをコードしている組換えポリヌクレオチドを含んでいる宿主細胞を準備すること;ならびに
ヘミセルロースを含んでいる培地において上記宿主細胞を培養することを包含しており、
上記宿主細胞は、上記組換えポリヌクレオチドを含んでいない細胞と比べて、より早く成長する、方法。 - 上記宿主細胞は、Saccharomyces sp.、Saccharomyces cerevisiae、Saccharomyces monacensis、Saccharomyces bayanus、Saccharomyces pastorianus、Saccharomyces carlsbergensis、Saccharomyces pombe、Kluyveromyces sp.、Kluyveromyces marxiamus、Kluyveromyces lactis、Kluyveromyces fragilis、Pichia stipitis、Sporotrichum thermophile、Candida shehatae、Candida tropicalis、Neurospora crassa、Zymomonas mobilis、Clostridium sp.、Clostridium phytofermentans、Clostridium thermocellum、Clostridium beijerinckii、Clostridium acetobutylicum、Moorella thermoacetica、Escherichia coli、Klebsiella oxytoca、Thermoanaerobacterium saccharolyticum、およびBacillus subtilisからなる群から選択される、請求項109に記載の方法。
- 上記宿主細胞は、上記組換えポリヌクレオチドと作動可能に連結されている誘導性のプロモータをさらに含んでいる、請求項109に記載の方法。
- 上記宿主細胞のヘミセルラーゼ活性は上記組換えポリヌクレオチドの発現によって促進される、請求項109に記載の方法。
- セルロースを分解する方法であって、
セルロースを含んでいる組成物を準備すること;および
上記組成物を、改変された生物から得られたセルラーゼ含有の酵素混合物と接触させることを包含しており、
セルラーゼを含有している上記酵素混合物は、改変されていない生物から得られたセルラーゼ含有の酵素混合物と比べて、低下したグルコシダーゼ活性を有しており、上記セルロースはセルラーゼ含有の上記酵素混合物によって分解される、方法。 - 上記生物はβ−グルコシダーゼをコードしている遺伝子の変異によって改変されている、請求項113に記載の方法。
- 上記生物はβ−グルコシダーゼの発現を低下させることによって改変されている、請求項113に記載の方法。
- 上記生物は真菌および細菌からなる群から選択される、請求項113〜115のいずれか1項に記載の方法。
- 上記生物は糸状菌である、請求項116に記載の方法。
- 上記セルロースは植物材料に由来する、請求項113〜117のいずれか1項に記載の方法。
- 上記植物材料は、スイッチグラス、Miscanthus、もみ殻、バガス、アマ、タケ、サイザル繊維、アバカ、麦わら、葉、刈り取った芝、トウモロコシの茎および葉、トウモロコシの穂軸、穀類の蒸留粕、マメ科の植物、サトウモロコシ、サトウキビ、テンサイの果肉、材木の切り屑、おが屑、ならびにバイオマス収穫物からなる群から選択される、請求項118に記載の方法。
Applications Claiming Priority (5)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US27183309P | 2009-07-24 | 2009-07-24 | |
| US61/271,833 | 2009-07-24 | ||
| US28552609P | 2009-12-10 | 2009-12-10 | |
| US61/285,526 | 2009-12-10 | ||
| PCT/US2010/043279 WO2011011796A2 (en) | 2009-07-24 | 2010-07-26 | Methods and compositions for improving sugar transport, mixed sugar fermentation, and production of biofuels |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| JP2013500019A true JP2013500019A (ja) | 2013-01-07 |
Family
ID=43497647
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2012521881A Pending JP2013500019A (ja) | 2009-07-24 | 2010-07-26 | 糖輸送、混合糖の発酵および生物燃料の産生を促進させる方法ならびに組成物 |
Country Status (13)
| Country | Link |
|---|---|
| US (4) | US8431360B2 (ja) |
| EP (1) | EP2456878A4 (ja) |
| JP (1) | JP2013500019A (ja) |
| KR (1) | KR20120089631A (ja) |
| CN (1) | CN102625844A (ja) |
| AU (1) | AU2010275388A1 (ja) |
| BR (1) | BR112012001664A2 (ja) |
| CA (1) | CA2765313A1 (ja) |
| EA (1) | EA201290068A1 (ja) |
| IN (1) | IN2012DN00345A (ja) |
| MX (1) | MX2012001071A (ja) |
| NZ (1) | NZ597001A (ja) |
| WO (1) | WO2011011796A2 (ja) |
Cited By (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| JP5689062B2 (ja) * | 2009-09-03 | 2015-03-25 | 国立大学法人京都大学 | 五炭糖輸送体 |
| WO2019044887A1 (ja) * | 2017-08-30 | 2019-03-07 | 国立研究開発法人理化学研究所 | 新規β-グルコシダーゼ、これを含む酵素組成物およびこれらを用いた糖液の製造方法 |
Families Citing this family (38)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| NZ597001A (en) * | 2009-07-24 | 2014-05-30 | Univ California | Methods and compositions for improving sugar transport, mixed sugar fermentation, and production of biofuels |
| US20110262983A1 (en) * | 2010-03-31 | 2011-10-27 | The United States Of America As Represented By The Secretary Of Agriculture | Metabolically engineered yeasts for the production of ethanol and other products from xylose and cellobiose |
| GB2488918B (en) | 2010-06-26 | 2014-03-05 | Virdia Ltd | Sugar mixtures and methods for production and use thereof |
| IL206678A0 (en) | 2010-06-28 | 2010-12-30 | Hcl Cleantech Ltd | A method for the production of fermentable sugars |
| IL207945A0 (en) | 2010-09-02 | 2010-12-30 | Robert Jansen | Method for the production of carbohydrates |
| US9096859B2 (en) | 2011-01-26 | 2015-08-04 | The Regents Of The University Of California | Microbial conversion of plant biomass to advanced biofuels |
| US20140057323A1 (en) | 2011-02-07 | 2014-02-27 | The Board Of Trustees Of The University Of Illinois | Enhanced cellodextrin metabolism |
| US9512495B2 (en) | 2011-04-07 | 2016-12-06 | Virdia, Inc. | Lignocellulose conversion processes and products |
| US9181563B2 (en) | 2011-04-11 | 2015-11-10 | Cargill, Incorporated | Compositions and methods for increased ethanol production from biomass |
| BR112013027197A2 (pt) * | 2011-04-22 | 2016-11-29 | Dsm Ip Assets Bv | célula de levedura capaz de converter açúcares incluindo arabinose e xilose |
| WO2013055785A1 (en) | 2011-10-10 | 2013-04-18 | Virdia Ltd | Sugar compositions |
| US20140342418A1 (en) * | 2011-10-17 | 2014-11-20 | The Board Of Trustees Ofthe University Of Illinois | Xylitol production from cellulosic biomass |
| WO2013155481A1 (en) | 2012-04-13 | 2013-10-17 | The Board Of Trustees Of The University Of Illinois | Improved cellodextrin transport and mixed sugar fermentation |
| GB2517338B (en) | 2012-05-03 | 2020-03-25 | Virdia Inc | A method for fractionating a liquid sample |
| CN103060390A (zh) * | 2012-08-29 | 2013-04-24 | 太仓市茂通化建有限公司 | 一种利用玉米秸秆水解液燃料丁醇的方法 |
| CN104140977B (zh) * | 2013-05-07 | 2018-10-23 | 中国科学院天津工业生物技术研究所 | Cdt-2的新用途以及利用其促进微生物细胞转运木寡糖的方法及其应用 |
| WO2015069796A1 (en) * | 2013-11-05 | 2015-05-14 | Board Of Regents, The University Of Texas System | Methods for engineering sugar transporter preferences |
| CN104844698B (zh) * | 2014-02-16 | 2020-09-25 | 中国科学院天津工业生物技术研究所 | 一种促进微生物细胞转运葡萄糖、木糖和阿拉伯糖的方法及其在生物基产品发酵中的应用 |
| JP6092157B2 (ja) * | 2014-06-09 | 2017-03-08 | 富士フイルム株式会社 | 電子機器、電子機器の表示制御方法、及び電子機器の表示制御プログラム |
| CN106460022B (zh) * | 2014-06-24 | 2018-06-05 | 加州大学董事会 | 木糖甙木糖醇寡聚物的生产方法 |
| WO2016012429A1 (en) * | 2014-07-24 | 2016-01-28 | Dsm Ip Assets B.V. | Yeast cell with improved pentose transport |
| JP6500406B2 (ja) * | 2014-12-01 | 2019-04-17 | セイコーエプソン株式会社 | 入出力制御装置、入出力制御プログラム |
| CN107108543A (zh) | 2015-01-07 | 2017-08-29 | 威尔迪亚公司 | 萃取和转化半纤维素糖的方法 |
| US10217619B2 (en) * | 2015-03-12 | 2019-02-26 | Thermo Finnigan Llc | Methods for data-dependent mass spectrometry of mixed intact protein analytes |
| BR112017025322A8 (pt) | 2015-05-27 | 2022-08-23 | Virdia Inc | Processos integrados para recuperação de hidrolisato celulósico após hidrólise de polpa de celulose |
| CN107236757A (zh) * | 2016-03-28 | 2017-10-10 | 中国科学院天津工业生物技术研究所 | 一种提高丝状真菌木质纤维素酶系表达及生物基化学品生产的方法 |
| WO2018027181A1 (en) | 2016-08-05 | 2018-02-08 | Kuehnle Agrosystems, Inc. | Producing and altering microbial fermentation products using non-commonly used lignocellulosic hydrolysates |
| CN109803672A (zh) | 2016-09-13 | 2019-05-24 | 阿勒根公司 | 稳定的非蛋白质梭菌毒素组合物 |
| CA3068461A1 (en) | 2017-06-30 | 2019-01-03 | Zimitech, Inc. | Engineered microorganisms for enhanced use of oligosaccharides |
| WO2019148192A1 (en) * | 2018-01-29 | 2019-08-01 | Novozymes A/S | Microorganisms with improved nitrogen utilization for ethanol production |
| ES2799298T3 (es) * | 2018-03-12 | 2020-12-16 | Indian Oil Corp Ltd | Sacarificación y co-fermentación simultánea en dos etapas para producir etanol a partir de lignocelulosa |
| CN110240271B (zh) * | 2018-04-28 | 2022-06-17 | 华东理工大学 | 一种处理含磷酸三乙酯废水的生物反应器的启动方法 |
| CN110982850B (zh) * | 2018-12-25 | 2022-07-29 | 华侨大学 | 一种半纤维素糖化能力增强的米曲霉工程菌合成木糖醇的方法 |
| CN109837232A (zh) * | 2019-03-27 | 2019-06-04 | 黑龙江大学 | 一种粗糙脉胞菌外切葡聚糖酶突变菌株的构建方法和应用 |
| CN111593014B (zh) * | 2020-06-24 | 2022-07-05 | 江南大学 | 联产1,3-丙二醇和d-1,2,4-丁三醇的方法 |
| CN112322563A (zh) * | 2020-10-20 | 2021-02-05 | 广西科学院 | 一种纤维素发酵工程菌及其构建方法和应用 |
| WO2022272119A2 (en) * | 2021-06-25 | 2022-12-29 | The Board Of Trustees Of The University Of Illinois | Synthetic toolkit for plant transformation |
| CN117821489A (zh) * | 2023-12-14 | 2024-04-05 | 湖北大学 | 一种利用重组酵母全细胞降解可再生塑料的方法 |
Family Cites Families (7)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US5100791A (en) * | 1991-01-16 | 1992-03-31 | The United States Of America As Represented By The United States Department Of Energy | Simultaneous saccharification and fermentation (SSF) using cellobiose fermenting yeast Brettanomyces custersii |
| EP1002059B1 (en) * | 1997-07-04 | 2007-12-26 | Novozymes A/S | Endo-beta-1,4-glucanases from saccharothrix |
| US7344876B2 (en) * | 2003-01-24 | 2008-03-18 | Phage Biotechnology, Inc. | Kluyveromyces strains metabolizing cellulosic and hemicellulosic materials |
| PT103331A (pt) * | 2005-08-05 | 2007-02-28 | Fundacao Da Faculdade De Cienc | Expressão dum transportador activo de xilose em saccharomyces cerevisiae modificada geneticamente |
| EP1994148B1 (en) * | 2005-11-16 | 2011-07-06 | Novozymes A/S | Polypeptides having endoglucanase activity and polynucleotides encoding same |
| WO2008155665A2 (en) * | 2007-05-09 | 2008-12-24 | University Of Stellenbosch | Method for enhancing cellobiose utilization |
| NZ597001A (en) * | 2009-07-24 | 2014-05-30 | Univ California | Methods and compositions for improving sugar transport, mixed sugar fermentation, and production of biofuels |
-
2010
- 2010-07-26 NZ NZ597001A patent/NZ597001A/en not_active IP Right Cessation
- 2010-07-26 CN CN2010800398589A patent/CN102625844A/zh active Pending
- 2010-07-26 CA CA2765313A patent/CA2765313A1/en not_active Abandoned
- 2010-07-26 KR KR1020127002889A patent/KR20120089631A/ko not_active Application Discontinuation
- 2010-07-26 JP JP2012521881A patent/JP2013500019A/ja active Pending
- 2010-07-26 WO PCT/US2010/043279 patent/WO2011011796A2/en active Application Filing
- 2010-07-26 EA EA201290068A patent/EA201290068A1/ru unknown
- 2010-07-26 IN IN345DEN2012 patent/IN2012DN00345A/en unknown
- 2010-07-26 US US12/843,844 patent/US8431360B2/en not_active Expired - Fee Related
- 2010-07-26 AU AU2010275388A patent/AU2010275388A1/en not_active Abandoned
- 2010-07-26 EP EP10803022A patent/EP2456878A4/en not_active Withdrawn
- 2010-07-26 BR BR112012001664A patent/BR112012001664A2/pt not_active IP Right Cessation
- 2010-07-26 MX MX2012001071A patent/MX2012001071A/es active IP Right Grant
-
2013
- 2013-03-13 US US13/802,533 patent/US8765410B2/en not_active Expired - Fee Related
-
2014
- 2014-05-28 US US14/289,570 patent/US9012177B2/en not_active Expired - Fee Related
-
2015
- 2015-01-28 US US14/608,018 patent/US20150299755A1/en not_active Abandoned
Non-Patent Citations (2)
| Title |
|---|
| JPN6014043238; Mol.Microbiol.,2005,55(1),p.90-103 * |
| JPN6014043241; Mol.Plant Pathol.,2008,9(2),p.203-11 * |
Cited By (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| JP5689062B2 (ja) * | 2009-09-03 | 2015-03-25 | 国立大学法人京都大学 | 五炭糖輸送体 |
| WO2019044887A1 (ja) * | 2017-08-30 | 2019-03-07 | 国立研究開発法人理化学研究所 | 新規β-グルコシダーゼ、これを含む酵素組成物およびこれらを用いた糖液の製造方法 |
Also Published As
| Publication number | Publication date |
|---|---|
| WO2011011796A2 (en) | 2011-01-27 |
| AU2010275388A1 (en) | 2012-01-12 |
| US20110020910A1 (en) | 2011-01-27 |
| WO2011011796A3 (en) | 2011-09-09 |
| KR20120089631A (ko) | 2012-08-13 |
| CN102625844A (zh) | 2012-08-01 |
| WO2011011796A8 (en) | 2011-12-22 |
| US8765410B2 (en) | 2014-07-01 |
| US9012177B2 (en) | 2015-04-21 |
| NZ597001A (en) | 2014-05-30 |
| US20150010985A1 (en) | 2015-01-08 |
| MX2012001071A (es) | 2012-10-09 |
| EP2456878A4 (en) | 2013-01-02 |
| IN2012DN00345A (ja) | 2015-05-08 |
| US8431360B2 (en) | 2013-04-30 |
| US20150299755A1 (en) | 2015-10-22 |
| AU2010275388A8 (en) | 2012-06-14 |
| US20130316437A1 (en) | 2013-11-28 |
| BR112012001664A2 (pt) | 2018-12-26 |
| CA2765313A1 (en) | 2011-01-27 |
| WO2011011796A9 (en) | 2011-06-23 |
| EA201290068A1 (ru) | 2013-05-30 |
| EP2456878A2 (en) | 2012-05-30 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| US9012177B2 (en) | Methods and compositions for improving sugar transport, mixed sugar fermentation, and production of biofuels | |
| EP2516663B1 (en) | Methods for improving the efficiency of simultaneous saccharification and fermentation reactions | |
| CA2689910A1 (en) | Compositions for degrading cellulosic material | |
| JP2010528624A (ja) | セルロース物質分解のための組成物 | |
| MX2012012171A (es) | Proceso para la produccion de celulas que tienen la capacidad de convertir arabinosa. | |
| EP2699589A2 (en) | Yeast cell capable of converting sugars including arabinose and xylose | |
| WO2013071112A1 (en) | A genetically modified strain of s. cerevisiae engineered to ferment xylose and arabinose | |
| WO2013155481A1 (en) | Improved cellodextrin transport and mixed sugar fermentation | |
| US11091753B2 (en) | Xylose fermenting yeast strains and processes thereof for ethanol production | |
| US20140154751A1 (en) | Enhanced fermentation of cellodextrins and beta-d-glucose | |
| US9670515B2 (en) | Methods for production of xylosylxylitol oligomers | |
| AU2012205191B2 (en) | Compositions for degrading cellulosic material | |
| Class et al. | Patent application title: METHODS AND COMPOSITIONS FOR IMPROVING SUGAR TRANSPORT, MIXED SUGAR FERMENTATION, AND PRODUCTION OF BIOFUELS | |
| BR112021012497A2 (pt) | Levedura recombinante e método para produzir etanol usando a mesma |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20130610 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20141014 |
|
| A02 | Decision of refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A02 Effective date: 20150310 |